Изобретение относится к области биотехнологии, в частности к определению 22FA серотипа Streptococcus pneumoniae с использованием полимеразной цепной реакции в режиме реального времени (ПЦР-РРВ) и может использоваться для идентификации возбудителей пневмококковых инфекций (ПИ) и оценки эффективности проводимых иммунопрофилактических мероприятий.
S. pneumoniae является грамположительным анаэробным видом бактерий. В зависимости от химических свойств полисахаридной капсулы выделяют около 50 серогрупп, образующих не менее 100 серотипов, многие из которых ассоциированы с инвазивными ПИ. Важным элементом эпидемиологического надзора за пневмококковыми инфекциями является микробиологический мониторинг, включающий антигенную и генетическую характеристику возбудителей ПИ, а также данные о чувствительности к антибиотикам. Антигенная характеристика возбудителей заключается в определении серотипов, что позволяет оценить эффективность существующих поливалентных вакцин. В России широкое применение получили 13-валентная конъюгированная пневмококковая вакцина (PCV13, «Превенар 13») и 23-валентная полисахаридная вакцина (PPV23, «Пневмовакс 23»). Определение спектра серотипов возбудителей ПИ позволяет планировать иммунопрофилактические мероприятия и оценивать их эффективность в группах лиц, вовлеченных в эпидемический процесс.
Для определения серотипов S. pneumoniae используются серологические способы, основанные на реакции набухания полисахаридной капсулы или реакции латекс-агглютинации. Широко используются и основанные на ПЦР способы детекции серотип-специфических последовательностей cps-локуса, такие как гены полисахаридной полимеразы wzy и флиппазы wzx, а также другие гены, участвующие в биосинтезе капсульного полисахарида (wcwV, galU, wciP, wzg) [Bentley, S.D. Genetic analysis of the capsular biosynthetic locus from all 90 pneumococcal serotypes / S.D. Bentley, D.M. Aanensen, A. Mavroidi et al. // PLoS Gene. - 2006. - Vol. 2. - №3. - P. 262-269. doi:10.1371/journal.pgen.0020031; Selva, L. Rapid and easy identification of capsular serotypes of Streptococcus pneumoniae by use of fragment analysis by automated fluorescence-based capillary electrophoresis / L. Selva, E. del Amo, P. Brotons et al. // J. Clin. Microbiol. - 2012. - Vol. 50. №11. P. 3451-3457. doi:10.1128/JCM.01368-12].
В 2006 году R. Pai и соавторы предложили алгоритм серотипирования S. pneumoniae, основанный на последовательных мультиплексных ПЦР [Pai, R. Sequential multiplex PCR approach for determining capsular serotypes of Streptococcus pneumoniae isolates / R. Pai, R.E. Gertz, B. Beall // J. Clin. Microbiol. - 2006. - Vol. 44. - №1. - P. 124-131. doi:10.1128/JCM.44.1.124-131.2006]. К недостаткам данного подхода относится необходимость использования электрофоретической детекции продуктов ПЦР в агарозном геле, что, наряду с другими недостатками включения данного этапа, может осложнять дифференциацию серотипов при проведении мультиплексной ПЦР.
Из уровня техники также известен способ определения 29 серотипов [Yun, K.W. Streptococcus pneumoniae type determination by multiplex polymerase chain reaction / K.W. Yun, E.Y. Cho, K.B. Hong et al. // Journal of Korean medical science. - 2011. - Vol. 26. - №8. - P. 971-978. doi:10.3346/jkms.2011.26.8.971]. Недостатком данного способа также является этап электрофореза.
Оптимальным способом одновременного определения нескольких серотип-специфических мишеней является ПЦР-РРВ с использованием флуоресцентно меченых зондов. Несмотря на преимущества ПЦР-РРВ для определения серотипов, эффективность ее применения не всегда очевидна, что обусловлено постоянной адаптацией возбудителей под давлением популяционного иммунитета, в том числе обусловленного иммунопрофилактикой с использованием поливалентных вакцин.
Центрами по контролю и профилактике заболеваний США (Centers for Disease Control and Prevention) рекомендованы последовательности олигонуклеотидов для определения серотипов S. pneumoniae с использованием ПЦР-РРВ и опубликованы алгоритмы проведения серотипирования для Латинской Америки, Африки, Азии и США [Real-time PCR Deduction of Pneumococcal Serotypes or Serogroups PCR based methods for identification of S. pneumoniae serotypes/serogroups. URL: www.cdc.gov/streplab/pneumococcus/resources.html? CDC_AA_refVal=https%3A%2F%2Fwww.cdc.gov%2Fstreplab%2Fpcr.html (дата обращения: 01.09.2021)]. Рекомендованные протоколы постановки ПЦР-РРВ предполагают определение трех серотип-специфических мишеней в одной реакции. Различия в способах серотипирования связаны с тем, что частота обнаружения тех или иных серотипов варьирует в зависимости от региона и параметров эпидемического процесса на каждой территории (доля инвазивных и неинвазивных форм ПИ, спектр циркулирующих серотипов и их изменение в зависимости от проводимых иммунопрофилактических мероприятий), которые определяют особенности проведения микробиологического мониторинга S. pneumoniae.
Детекция дополнительных серотип-специфических мишеней, также как и исключение из анализа мишеней для серотипов, не характерных для наблюдаемой территории, позволяет повысить долю определяемых серотипов и эффективность лабораторного исследования в целом. Уменьшение доли штаммов, нетипируемых с помощью стандартных серологических или основанных на ПЦР методик, способствует накоплению информации об эпидемиологических особенностях циркулирующих возбудителей для планирования и контроля эффективности программ по вакцинации. В связи с этим существует потребность в разработке олигонуклеотов для выявления 22FA серотипа S. pneumoniae: праймеров и зонда для проведения ПЦР-РРВ в мультиплексном формате.
Технический результат заявляемого изобретения направлен на выявление ДНК 22FA серотипа S. pneumoniae с высокой степенью специфичности в образцах биологического материала посредством таких олигонуклеотидов праймеров и флоуресцентного зонда, которые позволяют эффективно определять серотип-специфическую последовательность ДНК с использованием широко распространенных методик и доступных материалов.
Технический результат достигается за счет применения в ПЦР-РРВ синтезированных олигонуклеотидов для определения 22FA серотипа S. pneumoniae, имеющих следующий олигонуклеотидный состав:
прямой праймер 22FAF SEQ ID NO: 1;
обратный праймер 22FAR SEQ ID NO: 2;
флоуресцентный зонд 22FAZ - SEQ ID NO: 3.
Праймеры представляют собой последовательности олигонуклеотидов для амплификации серотип-специфического фрагмента ДНК, флоуресцентный зонд является олигонуклеотидом, содержащим флуорофор и гаситель флуоресценции, позволяющим детектировать амплифицированный фрагмент.
Заявляемые олигонуклеотиды разработаны на основе генов S. pneumoniae, входящих в cps-локус [Bentley, S.D. Genetic analysis of the capsular biosynthetic locus from all 90 pneumococcal serotypes / S.D. Bentley, D.M. Aanensen, A. Mavroidi et al. // PLoS Gene. 2006. Vol. 2. №3. P. 262-269. doi: 10.1371/journal.pgen.0020031]. С этой целью были проанализированы фрагменты последовательностей генома S. pneumoniae (cps-локуса) 22FA серотипа для определения консервативных участков ДНК, не имеющих гомологии с фрагментами cps-локуса представителей других серотипов S. pneumoniae. В результате проведенного анализа был выбран гомологичный для 22FA серотипа S. pneumoniae фрагмент cps-локуса, к которому были подобраны праймеры и зонд для амплификации фрагмента 77 пар оснований: прямой 22FAF - SEQ ID NO: 1; обратный 22FAR - SEQ ID NO: 2 и флоуресцентный зонд 22FAZ - SEQ ID NO: 3.
Заявляемое изобретение является результатом работы в рамках исследования российских штаммов S. pneumoniae, проделанной в ФБУН ЦНИИ Эпидемиологии Роспотребнадзора (Москва, Россия).
Для подбора последовательностей-мишеней - мест посадки олигонуклеотидов были использованы фрагменты референсных геномов 22FA серотипа S. pneumoniae, взятые из базы данных GenBank. Для поиска консервативных последовательностей применяют современные алгоритмы in silico анализа нуклеотидных последовательностей и программы, находящиеся в открытом доступе, включая Mega и Unipro UGENE, олигокалькуляторы (Oligo Calculators) Thermo Fisher Scientific. Составляют перечень консервативных участков, характерных только для 22FA серотипа S. pneumoniae, к которым подбирают олигонуклеотидные последовательности прямого и обратного праймеров, а также флоуресцентный зонд. Упомянутые олигонуклеотидные последовательности приведены в Таблице 1.
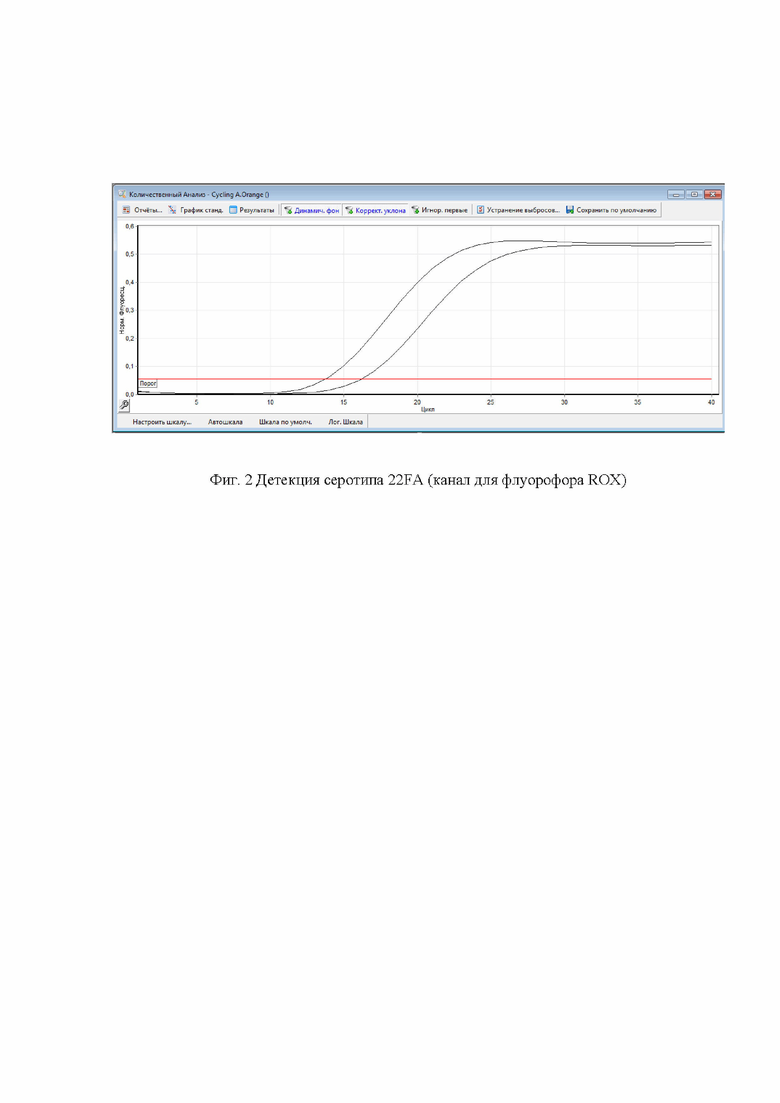
В качестве положительного внутреннего контроля используется фрагмент cps-локуса общего для всех серотипов SEQ ID NO: 4-6, с праймерами cpsA-f и cpsA-r [Pai, R. Sequential multiplex PCR approach for determining capsular serotypes of Streptococcus pneumoniae isolates / R. Pai, R.E. Gertz, B. Beall // J. Clin. Microbiol. 2006. Vol. 44. №1. - P.124-131. doi:10.1128/JCM.44.1.124-131.2006] и зондом cpsA-Z2 [Миронов, К.О. Методика ПЦР в режиме реального времени для определения серотипов Streptococcus pneumoniae / К.О. Миронов, А.Е. Платонов, Е.А. Дунаева и др. // Журнал микробиологии, эпидемиологии и иммунобиологии. 2014. №1. С. 41-48.]. Для детекции cps-положительных образцов используют канал для флуорофора Су5.5.
Анализ заявляемых последовательностей с использованием ресурса Primer BLAST продемонстрировал, что прямой 22FAF (SEQ ID NO: 1) и обратный 22FAR (SEQ ID NO: 2) праймеры амплифицируют консервативный для 15ВС серотипа участок со 100% специфичностью.
где ROX - флуорофор, BHQ2 - гаситель флуоресценции.
Заявляемое решение также поясняется:
на Фиг 1. на которой представлен консервативный участок генома 22FA серотипа S. pneumoniae и места посадки праймеров и флоуресцентного зонда, где:
1 - прямой праймер 22FAF (SEQ ID NO: 1);
2 - флоуресцентный зонд 22FAZ, (SEQ ID NO: 3);
3 - обратный праймер 22FAR, (SEQ ID NO: 2);
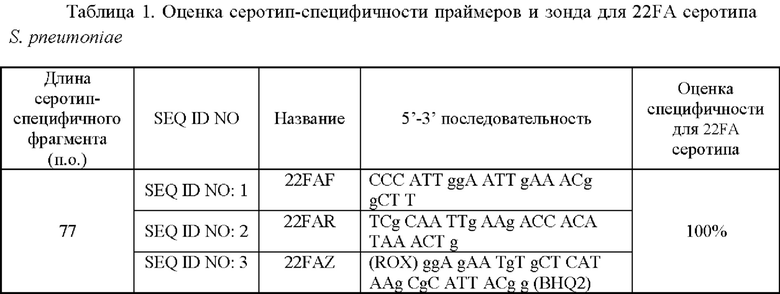
на Фиг. 2, где показана детекция серотипа 22FA (канал для флуорофора ROX);
на Фиг. 3, где показана детекция cps-положительных образцов (канал для флуорофора Су5.5)
В качестве биологического материала предпочтительно использовать спинномозговую жидкость или образцы культур S. pneumoniae.
Выделение ДНК из биологического материала или штаммов проводят в соответствии с МУ 1.3.2569-09 «Организация работ лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Выделение ДНК из биологического материала производят с помощью комплекта реагентов в соответствии с инструкцией производителя. Для выделения ДНК может быть использован комплект реагентов «РИБО-преп» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) или любой аналогичный набор для выделения ДНК. Оптимальная концентрация ДНК - 103-105 копий в 10 мкл.
ПЦР-РРВ проводится с применением заявляемых олигонуклеотидов - праймеров и зонда, для серотип-специфичной мишени 22FA серотипа, представленных в Таблице 1.
ПЦР-РРВ проводят при следующих условиях:
Общий объем реакционной смеси - 25 мкл.
Компоненты ПЦР смешиваются следующим образом:
(a) 10 мкл смеси, содержащей:
- олигонуклеотидные праймеры SEQ ID NO: 1, 2, 4, 5 - по 0,5 мкМ;
- флоуресцентный зонд SEQ ID NO: 3,6 - 0,2 мкМ;
- dNTPs - 0,44 мМ.
(b) реактив, содержащий рекомбинантный фермент Taq ДНК-полимеразы, например 0,5 мкл реактива «Полимераза TaqF» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) или любого аналогичного коммерческого набора в соответствии с инструкцией производителя.
(c) ПЦР-буфер, содержащий MgCl2, например 4,5 мкл ПЦР-буфера «ОТ-ПЦР-смесь-2 FEP/FRT» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) или любого аналогичного коммерческого набора в соответствии с инструкцией производителя.
(d) выделенная ДНК - 10 мкл.
ПЦР-РРВ проводят с флуоресцентной детекцией на приборе «Rotor-Gene Q» («Qiagen», Германия) или на любом другом приборе с 5 каналами детекции.
Амплификацию проводят по следующей программе: 1 цикл 95°С в течение 15 минут, 5 циклов при температуре 95°С - 5 секунд / 60°С - 20 секунд / 72°С - 5 секунд, затем 40 циклов при температуре 95°С - 5 секунд / 60°С - 20 сек / 72°С - 5 секунд. Детекция флуоресценции проводится на этапе 60°С по каналу для флуорофора ROX.
Для анализа результатов задают пороговую линию, соответствующую величине 8 10% от максимального сигнала положительного образца. Образцы, для которых кривые флуоресценции пересекают пороговую линию, и, при этом кинетика накопления флуоресцентного сигнала является экспоненциальной, являются положительными, то есть содержат серотип-специфическую мишень для 22FA серотипа S. pneumoniae.
Реализация заявляемого изобретения поясняется следующими примерами:
Пример 1. Получение олигонуклеотидов для определения 22FA серотипа S pneumoniae.
Для подбора последовательностей-мишеней - мест посадки олигонуклеотидов были использованы фрагменты референсных геномов 22FA серотипа S. pneumoniae, взятые из базы данных GenBank. Для поиска консервативных последовательностей были использованы современные алгоритмы in silico анализа нуклеотидных последовательностей и программы, находящиеся в открытом доступе, включая Mega и Unipro UGENE, олигокалькуляторы (Oligo Calculators) Thermo Fisher Scientific. Был составлен перечень консервативных участков, характерных только для 22FA серотипа S. pneumoniae, к которым были подобраны олигонуклеотидные последовательности прямого 22FAF и обратного 22FAR праймеров, а также флоуресцентный зонд 22FAZ.
В качестве положительного внутреннего контроля используется фрагмент cps-локуса общего для всех серотипов S. pneumoniae SEQ ID NO: 4-6.
Анализ упомянутых последовательностей с использованием ресурса Primer BLAST показал, что прямой 22FAF и обратный 22FAR праймеры амлифицируют консервативный для 22FA серотипа участок со 100% специфичностью.
Олигонуклеотиды для определения 22FA серотипа S. pneumoniae - прямой праймер 22FAF, обратный праймер 22FAR, флоуресцентный зонд 22FAZ, представлены уникальными последовательностями SEQ ID NO: 1-3 соответственно.
Пример 2: Детекция 22FA серотипа S. pneumoniae методом ПЦР-РРВ.
Определение 22FA серотипа S. pneumoniae проводят проводят методом ПЦР-РРВ при следующих условиях:
Общий объем реакционной смеси - 25 мкл. Компоненты ПЦР смешиваются следующим образом:
(a) 10 мкл смеси, содержащей:
- олигонуклеотидные праймеры SEQ ID NO: 1, 2, 4, 5 по 0,5 мкМ;
- флоуресцентный зонд SEQ ID NO: 3,6 - 0,2 мкМ;
- dNTPs - 0,44 мМ.
(b) 0,5 мкл реактива «Полимераза TaqF» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия).
(c) 4,5 мкл ПЦР-буфера «ОТ-ПЦР-смесь-2 FEP/FRT» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия).
(d) выделенная ДНК - 10 мкл.
ПЦР-РРВ проводили с флуоресцентной детекцией на приборе с 5 каналами детекции - «Rotor-Gene Q» («Qiagen», Германия).
Подготовленный описанным способом материал, содержащий уникальные олигонуклеотидные последовательности SEQ ID NO: 1-3 используют для определения 22FA серотипа S. pneumoniae в биологических образцах.
Пример 3. Обнаружение 22FA серотипа S. pneumoniae в образцах биологического материала.
Для определения 22FA серотипа S. pneumoniae было выбрано 47 смывов бактериальных культур. Выделение ДНК из биологического материала проводили в соответствии с МУ 1.3.2569-09 «Организация работ лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для выделения ДНК использовался набор реагентов «РИБО-преп» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия), концентрация ДНК 103 копий в 10 мкл.
ПЦР-РРВ проводили в условиях, описанных в Примере 2, с использованием уникальных олигонуклеотидных последовательностей SEQ ID NO: 1-3.
Амплификацию проводили по следующей программе: 1 цикл 95°С в течение 15 минут, 5 циклов при температуре 95°С - 5 секунд / 60°С - 20 секунд / 72°С - 5 секунд, затем 40 циклов при температуре 95°С - 5 секунд / 60°С - 20 сек / 72°С - 5 секунд. Детекцию флуоресценции проводили на этапе 60°С по каналу для флуорофора ROX. Для анализа результатов задали пороговую линию, соответствующую величине 10% от максимального сигнала положительного образца. Для двух образцов кривые флуоресценции пересекли пороговую линию и при этом кинетика накопления флуоресцентного сигнала была экспоненциальной, что свидетельствует о том, что данные образцы содержат серотип-специфическую мишень 22FA серотипа S. pneumoniae.
Таким образом, из всей выборки было выявлено два образца, принадлежащих к 22FA серотипу S. pneumoniae. Принадлежность данных образцов к 22FA серотипу S. pneumoniae подтверждена анализом данных полногеномного секвенирования с использованием программ «SeroBA» и «PneumoCaT».
Пример 4. Обнаружение 22FA серотипа S. pneumoniae в образцах биологического материала.
Для определения 22FA серотипа S. pneumoniae было выбрано 30 смывов с бактериальных культур. Выделение ДНК из биологического материала проводили в соответствии с МУ 1.3.2569-09 «Организация работ лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для выделения ДНК использовался набор реагентов «РИБО-преп» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия), концентрация ДНК 105 копий в 10 мкл.
ПЦР-РРВ проводили в условиях, описанных в Примере 2, с использованием уникальных олигонуклеотидных последовательностей SEQ ID NO: 1-3.
Амплификацию проводили по следующей программе: 1 цикл 95°С в течение 15 минут, 5 циклов при температуре 95°С - 5 секунд / 60°С - 20 секунд / 72°С - 5 секунд, затем 40 циклов при температуре 95°С - 5 секунд / 60°С - 20 сек / 72°С - 5 секунд. Детекцию флуоресценции проводили на этапе этапе 60°С по каналу для флуорофора ROX. Для анализа результатов задали пороговую линию, соответствующую величине 8% от максимального сигнала положительного образца. Для одного образца кривая флуоресценции пересекла пороговую линию и при этом кинетика накопления флуоресцентного сигнала была экспоненциальной, что свидетельствует о том, что данный образец содержит серотип-специфическую мишень для 22FA серотипа S. pneumoniae.
Таким образом, из всей выборки был выявлен один образец, принадлежащий к 22FA серотипу S. pneumoniae. Принадлежность данного образца к 22FA серотипу S. pneumoniae подтверждена анализом данных полногеномного секвенирования с использованием программ «SeroBA» и «PneumoCaT».
Пример 5. Обнаружение 22FA серотипа S. pneumoniae в образцах биологического материала.
Для определения 22FA серотипа S. pneumoniae было выбрано 45 смывов бактериальных культур. Выделение ДНК из биологического материала проводили в соответствии с МУ 1.3.2569-09 «Организация работ лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для выделения ДНК использовался набор реагентов «РИБО-преп» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия), концентрация ДНК 103 копий в 10 мкл.
ПЦР-РРВ проводили в условиях, описанных в Примере 2, с использованием уникальных олигонуклеотидных последовательностей SEQ ID NO: 1-3.
Амплификацию проводили по следующей программе: 1 цикл 95°С в течение 15 минут, 5 циклов при температуре 95°С - 5 секунд / 60°С - 20 секунд / 72°С - 5 секунд, затем 40 циклов при температуре 95°С - 5 секунд / 60°С - 20 сек / 72°С - 5 секунд. Детекцию флуоресценции проводили на этапе этапе 60°С по каналу для флуорофора ROX. Для анализа результатов задали пороговую линию, соответствующую величине 9% от максимального сигнала положительного образца. Кривая флуоресценции не пересекла пороговую линию, что свидетельствовало о том, что в выборке отсутствуют образцы, содержащие серотип-специфическую мишень для 22FA серотипа S. pneumoniae. Принадлежность образцов к другим серотипам S. pneumoniae подтверждена анализом данных полногеномного секвенирования с использованием программ «SeroBA» и «PneumoCaT».
Таким образом, заявляемое изобретение позволяет выявлять в биологических образцах ДНК 22FA серотипа S. pneumoniae. Синтезированные олигонуклеотиды SEQ ID NO: 1-3 не дают перекрестных реакций с другими протестированными серотипами S. pneumoniae, амлифицируют консервативный для 22FA серотипа участок со 100% специфичностью и позволяют определять наличие или отсутствие в образцах биологического материала серотип-специфической мишени для 22FA серотипа S. pneumoniae.
--->
Перечень последовательностей
<110> ФБУН ЦНИИ Эпидемиологии Роспотребнадзора
<120> Олигонуклеотиды для определения 22FA серотипа Streptococcus pneumoniae
<160> NUMBER OF SEQ ID NO: NOS: 6
<210> SEQ ID NO: NO 1
<211> 22
<212> DNA
<213> Streptococcus pneumoniae
<400> SEQUENCE 1 (22FAF):
CCC ATT ggA ATT gAA ACg gCT T 22
<210> SEQ ID NO: NO 2
<211> 25
<212> DNA
<213> Streptococcus pneumoniae
<400> SEQUENCE 2 (22FAR):
TCg CAA TTg AAg ACC ACA TAA ACT g 25
<210> SEQ ID NO: NO 3
<211> 28
<212> DNA
<213> Streptococcus pneumoniae
<400> SEQUENCE 3 (22FAZ):
(ROX) ggA gAA TgT gCT CAT AAg CgC ATT ACg g (BHQ2) 28
<210> SEQ ID NO: NO 4
<211> 27
<212> DNA
<213> Streptococcus pneumoniae
<400> SEQUENCE 4 (cpsА-f):
GCA GTA CAG CAG TTT GTT GGA CTG ACC 27
<210> SEQ ID NO: NO 5
<211> 26
<212> DNA
<213> Streptococcus pneumoniae
<400> SEQUENCE 5 (cpsА-r):
GAA TAT TTT CAT TAT CAG TCC CAG TC 26
<210> SEQ ID NO: NO 6
<211> 29
<212> DNA
<213> Streptococcus pneumoniae
<400> SEQUENCE 6 (cpsА-Z2):
(Cy5.5)GTG CTG TCA CAC TCG TCA GTT GCG TAA CA(BHQ3)29
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Олигонуклеотиды для определения 12FAB серотипа Streptococcus pneumoniae | 2021 |
|
RU2795021C1 |
| Олигонуклеотиды для определения 15BC серотипа Streptococcus pneumoniae | 2021 |
|
RU2795316C1 |
| Олигонуклеотиды для определения 8 серотипа Streptococcus pneumoniae | 2021 |
|
RU2802081C2 |
| Мультиплексная ПЦР-смесь для определения серотипов 12FAB, 15BC, 22FA, 8 Streptococcus pneumoniae и способ ее применения | 2021 |
|
RU2787181C1 |
| Набор олигонуклеотидных праймеров и зондов для определения аллей полиморфизма rs55986091 и способ его применения | 2022 |
|
RU2804110C1 |
| Олигонуклеотиды для определения мутации S:Ins214EPE SARS-CoV-2 | 2022 |
|
RU2795017C1 |
| Способ генотипирования гена TLR8 по полиморфизму rs3764880 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805864C1 |
| Способ генотипирования гена TLR2 по полиморфизму rs5743708 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805861C1 |
| Способ генотипирования гена TLR2 по полиморфизму rs3804100 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805860C1 |
| Способ генотипирования гена TLR1 по полиморфизму rs5743551 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805859C1 |

Изобретение относится к области биотехнологии, в частности к набору праймеров и зонда для определения 22FA серотипа Streptococcus pneumoniae методом полимеразной цепной реакции, проводимой в режиме реального времени. Олигонуклеотиды, входящие в состав набора, не дают перекрестных реакций с другими протестированными серотипами S. pneumoniae, амплифицируют консервативный для 22FA серотипа участок со 100% специфичностью и позволяют определять наличие или отсутствие в образцах биологического материала серотип-специфической мишени для 22FA серотипа S. pneumoniae. 2 з.п. ф-лы, 3 ил., 1 табл., 5 пр.
1. Набор праймеров и зонда для определения 22FA серотипа Streptococcus pneumoniae методом полимеразной цепной реакции, проводимой в режиме реального времени, и имеющих следующий нуклеотидный состав:
прямой праймер (22FAF) - SEQ ID NO: 1;
обратный праймер (22FAR) - SEQ ID NO: 2;
флоуресцентный зонд (22FAZ) - SEQ ID NO: 3.
2. Набор праймеров и зонда по п. 1, где прямой и обратный праймеры комплементарны участку гена S. pneumoniae 22FA серотипа, не имеющего гомологии с фрагментами cps-локуса представителей других серотипов S. pneumoniae.
3. Набор праймеров и зонда по п. 1, где флоуресцентный зонд является олигонуклеотидом, содержащим флуорофор ROX и гаситель флуоресценции BHQ2, позволяющим детектировать амплифицированный фрагмент.
| KI WOOK YUN et al., Streptococcus pneumoniae Type Determination by Multiplex Polymerase Chain Reaction, J Korean Med Sci, 2011, 26(8), pp.971-978 | |||
| REKHA PAI et al., Sequential Multiplex PCR Approach for Determining Capsular Serotypes of Streptococcus pneumoniae Isolates, J Clin Microbiol, 2006, 44(1), pp | |||
| Аппарат для радиометрической съемки | 1922 |
|
SU124A1 |
| К.О | |||
| МИРОНОВ и др., | |||
Авторы
Даты
2023-08-22—Публикация
2021-12-24—Подача