Изобретение относится к ветеринарной микробиологии, в частности к лабораторной диагностике возбудителей инфекционных заболеваний, а именно к средствам диагностики инфекции у животных с помощью полимеразной цепной реакции.
Известно в медицине проведение с помощью препаратов диагностику криптоспоридиоза посредством гистологического исследования биоптата, дофиксацию срезов метанолом, экспозицию при окраске карболовым фуксином осуществляют в течение 5-10 мин, в качестве деклорайзера применяют 5% серную кислоту. Известное техническое решение обеспечивает положительный эффект в виде выявления объективных критериев диагностики криптоспоридиоза, обеспечивающих тактику лечения (патент РФ №2229128, кл. G01N 1/30, 2004 г.).
Наиболее близким по технической сущности является изобретение по патенту RU №2726242, кл. C12Q 1/68, 2020 г., включающее буфер для проведения полимеразной цепной реакции, смесь для ее проведения, состоящая из дезоксинуклеозидтрифосфатов, олигонуклеотидных праймеров и флуоресцентных зондов специфичные для участка генома животного и для внутреннего контрольного образца; смесь ферментов из ДНК полимеразы с антителами, ингибирующих активность фермента, TAQ POLYMERASE, внутренний контрольный образец в виде суспензии бактериофага Т4 с концентрацией 5×105 фаговых частиц на 1 мкл, отрицательный контрольный образец, представляющий собой смесь рекомбинантных плазмидных ДНК, содержащих фрагмент генома животного и фрагмент генома бактериофага Т4 с нуклеотидной последовательностью:
T4F: 5`-TACATATAAATCACGCAAAGC-3` - прямой праймер
T4R: 5`-TAGTATGGCTAATCTTATTGG-3` - обратный праймер
Т4Р: СУ5-5` - ACATTGGCACTGACCGAGTTC-3`-BHQ1 - зонд, взятых в объемном соотношении 1:1.
Недостатком известного технического решения является отсутствие возможности выявления ДНК возбудителя криптоспоридиоза.
(Cryptosporidiosis) в биологическом материале животных, в воде водоемов и кормах.
Техническим результатом является расширение функциональных возможностей и повышение точности при выявлении возбудителя криптоспоридиоза (Cryptosporidiosis).
Технический результат достигается тем, что в тест-системе для выявления ДНК возбудителя криптоспоридиоза (Cryptosporidiosis) в биологическом материале животных, в воде из водоемов и кормах с помощью полиме-разной цепной реакции в режиме реального времени, включающей буфер для проведения полимеразной цепной реакции, смесь для ее проведения, состоящая из дезоксинуклеозидтрифосфатов, олигонуклеотидных праймеров и флуоресцентных зондов специфичные для участка генома животного и для внутреннего контрольного образца; смесь ферментов из ДНК полимеразы с антителами, ингибирующих активность фермента, TAQ POLYMERASE, внутренний контрольный образец в виде суспензии бактериофага Т4 с концентрацией 5×103 фаговых частиц на 1 мкл, отрицательный контрольный образец, представляющий собой смесь рекомбинантных плазмидных ДНК, содержащих фрагмент генома животного и фрагмент генома бактериофага Т4 с нуклеотидной последовательностью:
T4F: 5`-TACATATAAATCACGCAAAGC-3` - прямой праймер
T4R: 5`-TAGTATGGCTAATCTTATTGG-3` - обратный праймер
Т4Р: СУ5-5`-ACATTGGCACTGACCGAGTTC-3`-BHQ1 - зонд, взятых в объемном соотношении 1:1, согласно изобретению для положительного контрольного образца используют фрагмент генома возбудителя криптоспоридиоза (Cryptosporidiosis) со следующей нуклеотидной последовательностью:
Crypto-F: 5`-AAGGGTTGTATTTATTAGATAA-3`
Crypto-R: 5`-GTCAGAAACTTGAATGATA-3`
Crypto-Z: НЕХ-5`-TCCGTAAAGTTATTATGAGTCACCAAT-3`-BHQ1,
при этом для ДНК возбудителя криптоспоридиоза (Cryptosporidiosis) используют флуоресцентный краситель - JOE(HEX)/Yellow.
Новизна заявляемой тест-системы состоит в идентификации ДНК возбудителя криптоспоридиоза (Cryptosporidiosis)c помощью полимеразной цепной реакции (ПЦР) с флуоресцентной детекцией в режиме реального времени с использованием специфичных для участка генома олигонуклеотидных праймеров флуоресцентно-меченного зонда. Такая постановка ПЦР в реальном времени сокращает и упрощает процедуру анализа, снижает риск контаминации. Кроме того, флуоресцентная детекция продуктов амплификации осуществляется с использованием принципа выщепления флуоресцентной метки на 5' конце олигонуклеотидного зонда.
Признаки, отличающие заявляемое техническое решение от прототипа, направлены на достижение технического результата и не выявлены при изучении данной и смежной областей науки и техники и, следовательно, соответствуют критерию «изобретательский уровень».
Заявляемую тест-систему рекомендовано использовать в специализированных ветеринарных, санитарно-эпидемиологических, животноводческих, сельскохозяйственных предприятиях, что соответствует критерию «промышленная применимость».
Тест-систему для выявления ДНК возбудителя криптоспоридиоза (Cryptosporidiosis) в биологическом материале животных с помощью полимеразной цепной реакции в режиме реального времени используют следующим образом.
Предварительно выделяют ДНК из биологического материала животного сорбционным методом. В качестве биологического материала по выбору может быть использованы: вода из водоемов, корма, фекалии, паренхиматозные органы, кишечник и тонкой кишки, мясо и сырые мясные продукты, сырое молоко, которые подвергают определенной обработке.
Проводят полимеразную цепную реакцию с флуоресцентной детекцией с проведением 40 циклов амплификации в реальном времени с использованием специфичных для участка генома ДНК возбудителя криптоспоридиоза (Cryptosporidiosis) олигонуклеотидных праймеров, зондов, флуоресцентных красителей: для специфического сигнала для ДНК возбудителя криптоспоридиоза (Cryptosporidiosis) используют флуоресцентный краситель - JOE(HEX)/Yellow и для внутреннего контрольного образца бактериофаг Т4 с концентрацией 5×103 фаговых частиц на 1 мкл - FAM/Green. Для положительного контрольного образца используют смесь, содержащую в одинаковом объемном соотношении фрагменты геномов возбудителя криптоспоридиоза (Cryptosporidiosis) и бактериофага Т4 со следующими нуклеотидными последовательностями:
Crypto-F: 5`-AAGGGTTGTATTTATTAGATAA-3`
Crypto-R: 5`-GTCAGAAACTTGAATGATA-3`
Crypto-Z: HEX-5`-TCCGTAAAGTTATTATGAGTCACCAAT-3`-BHQ1,
T4F: 5`-TACATATAAATCACGCAAAGC-3` - прямой праймер
T4R: 5`-TAGTATGGCTAATCTTATTGG-3` - обратный праймер
T4P: FAM-5`-ACATTGGCACTGACCGAGTTC-3`-BHQ1 - зонд.
Затем измеряют накопления флуоресцентных сигналов по каналам соответствующих флуоресцентных красителей, проводят интерпретацию результатов на основании наличия или отсутствия пересечения кривой флуоресценции с пороговой линией, если кривые накопления флуоресцентного сигнала выходят до 35 цикла, то результат реакции считается положительным, а если кривые не пересекают пороговую линию или пересекают ее после 35 цикла, то результат реакции - отрицательный.
Для повышения точности идентификации возбудителя криптоспоридиоза (Cryptosporidiosis) для внутреннего контрольного образца используют бактериофаг Т4 с концентрацией 5×103 фаговых частиц на 1 мкл, если концентрация фаговых частиц отклоняется в большую или меньшую сторону, то наблюдаются повторности сомнительных образцов. Для положительного контрольного образца используют смесь, содержащую фрагменты геномов нативного бактериофага Т4 и ДНК возбудителя криптоспоридиоза (Cryptosporidiosis) взятых в соотношении 1:1, со следующими нуклеотидны-ми последовательностями:
Crypto-F: 5`-AAGGGTTGTATTTATTAGATAA-3`
Crypto-R: 5`-GTCAGAAACTTGAATGATA-3`
Crypto-Z: НЕХ-5`-TCCGTAAAGTTATTATGAGTCACCAAT-3`-BHQ1,
T4F: 5`-TACATATAAATCACGCAAAGC-3` - прямой праймер
T4R: 5`-TAGTATGGCTAATCTTATTGG-3` - обратный праймер
Т4Р: FAM-5`-ACATTGGCACTGACCGAGTTC-3`-BHQ1 - зонд.
При конструировании праймеров и зонда основными требованиями были: степень гомологии (комплементарность) с выбранным участком гена; отсутствие самокоплементарных участков внутри олигонуклеотидов и комплементарности друг другу, чтобы не допускать возникновения устойчивых вторичных структур (димеров); близость значений температуры отжига праймеров.
Конструирование специфических праймеров и зонда осуществляли с помощью компьютерных программ на основании анализа нуклеотидных последовательностей референтных штаммов и изолятов, опубликованных на ресурсе GenBank и подбора условий для проведения ПЦР в реальном времени с применением разработанных праймеров и зонда, несущего флуорофор и тушитель, и комплементарного части амплифицируемого со специфическими праймерами фрагмента.
Праймеры, специфичные для генома криптоспоридий (Cryptosporidium spp.) были отобраны на основе нуклеотидной последовательности гена 18S rRNA генома (LOCUS AF093490 1746 bp DNA linear INV04-MAY-2010 DEFINITION Cryptosporidium parvum strain Bovine C. parvum genotype (BOH6) small subunit ribosomal RNA gene, complete sequence.)
Дизайн олигонуклеотидных праймеров разработан с использованием специализированного программного обеспечения Primer Express Software v3.0 (Applied Biosystems). В результате анализа выбран участок между 100 и 400 нуклеотидами, содержащий уникальные нуклеотидные последовательности. С помощью программы «Oligo 6.0» рассчитаны первичные структуры олигонуклеотидных праймеров, фланкирующих выбранный участок генома.
Crypto-F: 5`-AAGGGTTGTATTTATTAGATAA-3`
Crypto-R: 5`-GTCAGAAACTTGAATGATA-3`
Специфичность праймеров (Crypto-F и Crypto-R) оценивали на сервере NCBI с использованием программы BLAST.
Для детекции продуктов амплификации был подобран олигонуклеотидный флуоресцентно-меченный зонд Crypto-Z (гомологичный участку нуклеотидной последовательности, ограниченной позициями отжига праймеров Crypto-F и Crypto-R). 3`-конец олигонуклеотидного зонда был помечен флуоресцентным красителем HEX, а 5`-конец зонда содержит гаситель флуоресценции BHQ1:
Crypto-Z: НЕХ-5`-TCCGTAAAGTTATTATGAGTCACCAAT-3`-BHQ1
Используя программу "Oligo 6.0" описаны основные свойства рассчитанных олигонуклеотидов, определившие возможность их использования в ПЦР в качестве праймеров и зонда. Ни одна из выбранных последовательностей не обнаружена в геномах других представителей кокцидий из группы простейших, а также в геномах любых видов животных и человека.
Для детекции продуктов амплификации подобран олигонуклеотидный флуоресцентно-меченный зонд Т4Р, комплементарный участку нуклеотидной последовательности, ограниченной позициями отжига праймеров T4F и T4R. Зонд был помечен красителем FAM. Используя программу "Oligo 6.0" описаны основные свойства рассчитанных олигонуклеотидов, определившие возможность их использования в ПЦР.
В качестве внутреннего контроля использовался бактериофаг Т4, имеющий геномную ДНК порядка 170 тысяч пар нуклеотидов (Enterobacteria phage T4T, complete genome GenBank: HM137666.1). В результате анализа был выбран участок между 400 и 600 нуклеотидами, содержащий уникальные нуклеотидные последовательности, рассчитаны первичные структуры олигонуклеотидных праймеров, фланкирующих выбранный участок генома. Праймеры были спроектированы с использованием Primer Express Software v3.0 (Applied Biosystems) и исследованы с использованием BLAST, чтобы подтвердить их специфичность.
Для детекции продуктов амплификации подобран олигонуклеотидный флуоресцентно-меченный зонд Т4Р, комплементарный участку нуклеотид-ной последовательности, ограниченной позициями отжига праймеров T4F и T4R. Зонд был помечен красителем FAM (Карбоксифлуоресцеин - длина волны возбуждения - 490 нм, флуоресценции - 520 нм). Используя программу "Oligo 6.0" описаны основные свойства рассчитанных олигонуклеотидов, определившие возможность их использования в ПЦР.
T4F TACATATAAATCACGCAAAGC прямой праймер
T4R TAGTATGGCTAATCTTATTGG обратный праймер
Т4Р FAM-5`-ACATTGGCACTGACCGAGTTC зонд.
Пример конкретного использования тест-системы для выявления ДНК возбудителя криптоспоридиоза (Cryptosporidiosis) с помощью полимеразной цепной реакции в режиме реального времени (ПЦР РВ)
Пример включает следующие этапы:
1. Отбор материала для исследования
Для исследования используют следующий материал:
• Мазки и соскобы слизистых оболочек носа. Снимают с помощью стерильного зонда, зонд помещают в пластиковую микропробирку объемом 1,5 мл с 0,5 мл стерильного физиологического раствора;
• Фрагменты паренхиматозных органов (фрагменты печени, легких, селезенки, а также миндалины, лимфоузлы и др.). Отбирают в стерильный контейнер.
• Цельная кровь. Кровь забирается в пробирку с 6% ЭДТА из расчета 50 мкл раствора ЭДТА на 1 мл крови, закрытую пробирку с кровью несколько раз переворачивают.
• Молоко, отбирают в объеме 10-30 мл в стерильную посуду.
• Отбор исследуемых образцов кормов/сырья проводят по национальным стандартам, устанавливающим порядок отбора проб для однородных групп сырья, пищевых продуктов и кормов.
2. Подготовка исследуемого материала
Мазки и соскобы со слизистых конъюнктивы в физиологическом растворе исследуют без предварительной подготовки.
Пробы тканей и органов, а также пробы продуктов животного происхождения гомогенизируют с использованием стерильных фарфоровых ступок и пестиков, затем обычно готовят 10% суспензию на стерильном физиологическом растворе или фосфатном буфере. Суспензию переносят в пробирку объемом 1,5 мл и центрифугируют при 10-12 тыс. об/мин в течение 2 минут. Надосадочную жидкость используют для экстракции НК. В некоторых случаях образовавшуюся суспензию не центрифугируют, а отстаивают при комнатной температуре в течение 5 мин, затем верхнюю фазу по 0,4-0,5 мл переносят пастеровской пипеткой (или наконечником с аэрозольным барьером) в пробирки на 1,5 мл, и в количестве 0,1 мл используют для выделения нуклеиновой кислоты (НК).
Молоко в объеме до 10 мл обеззараживают и центрифугируют при 3 тыс об/мин в течение 10-15 мин. Надосадочную жидкость осторожно отбирают, оставив над осадком примерно 0,2 мл жидкости. Осадок ресуспендируют в оставшейся надосадочной жидкости и 0,1 мл суспензии используют для выделения ДНК.
От кормов/сырья плотной консистенции отбирают на исследование общую пробу весом 10-50 г. Гранулированные или консервированные корма/сырье перед исследованием (10-20 г) растирают в ступке до гомогенного состояния. Отбирают лабораторные пробы (20-40 мг) в одноразовые микропробирки вместимостью 1,5 мл и направляют на выделение НК.
3. Проведение анализа
Исследование проводили с помощью набора реагентов «ПЦР-КРИПТОСПОРИДИОЗ-ФАКТОР» для выявления ДНК возбудителя криптоспоридиоза (Cryptosporidium spp.)». Набор состоит из комплекта реагентов для проведения мультиплексной ПЦР (Таблица 1, комплект №1) и комплекта контрольных образцов (Таблица 2, комплект №2). Набор выпускается в двух вариантах: 1) Для анализа 55 образцов (включая контрольные образцы)
2) Для анализа 110 образцов (включая контрольные образцы).
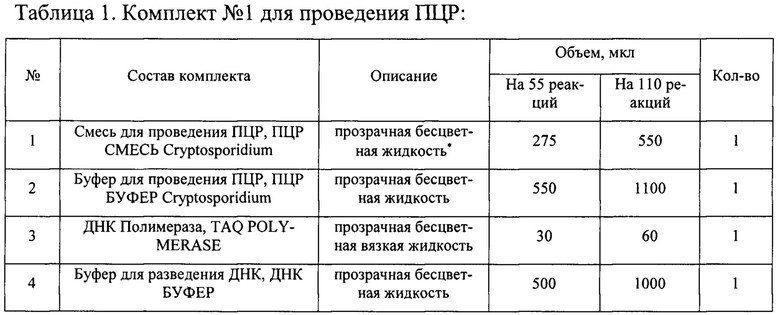
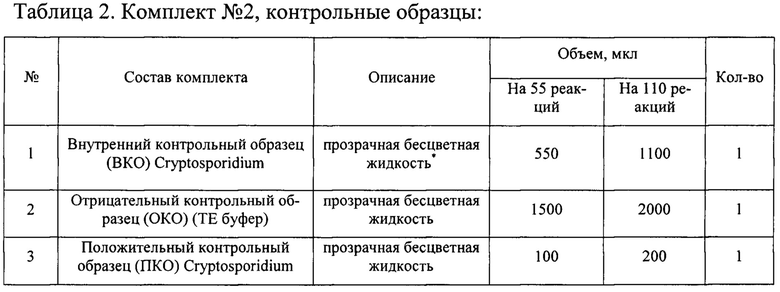
Наборы используют в соответствии с инструкцией по применению набора реагентов «ПЦР-КРИПТОСПОРИДИО3-ФАКТОР» для выявления ДНК возбудителя криптоспоридиоза (Cryptosporidium spp.) в биологическом материале и кормах методом полимеразной цепной реакции (ПЦР) с флуоресцентной детекцией в режиме реального времени, 21.10.60-221-51062356-2020 Для диагностики in vitro, http://www.vetfaktor.ru
Исследование с помощью набора реагентов «ПЦР - КРИПТОСПОРИДИОЗ-ФАКТОР» состоит из трех циклов:
• экстракция ДНК;
• проведение ПЦР РВ;
• учет результатов анализа.
4. Экстракция (выделение) НК из клинического материала
Отобрать необходимое количество одноразовых пробирок объемом 1,5 мл, включая отрицательный контроль выделения. Внести во все пробирки с исследуемыми образцами, включая пробирку для ОКО, по 10 мкл ВКО Cryptosporidium.
Внести исследуемые пробы в объеме согласно инструкции к набору для выделения ДНК, в пробирку отрицательного контроля выделения вместо исследуемой пробы внести ОКО (пробирку обозначить как ВК-).
Выделять ДНК из анализируемых и контрольных образцов согласно протоколу инструкции производителя набора для выделения ДНК.
Выделенную ДНК можно хранить в течение одной недели при температуре от 2°С до 8°С или в течение года при температуре не выше минус 16°С.
5. Подготовка образцов к проведению ПЦР
Общий объем реакционной смеси - 25 мкл, объем ДНК-пробы - 10 мкл.
Успешное прохождение реакции контролируют использованием ПКО Cryptosporidium, ВКО Cryptosporidium и ДНК буфера.
В отдельной пробирке смешать компоненты набора из расчета на каждую реакцию ПЦР:
• 5 мкл ПЦР СМЕСЬ Cryptosporidium
• 10 мкл ПЦР БУФЕР Cryptosporidium
• 0,5 мкл TAQ POLYMERASE
Перемешать смесь на вортексе и сбросить капли кратковременным центрифугированием.
Отобрать необходимое количество пробирок для амплификации ДНК исследуемых и контрольных проб. Внести по 15 мкл приготовленной реакционной смеси.
Используя наконечники с фильтром в подготовленные пробирки внести:
а) в пробирку отрицательного контроля ПЦР (К-) 10 мкл ДНК буфера;
б) в ряд пробирок для исследуемых проб - в каждую внести по 10 мкл ДНК соответствующей пробы, полученной по п. 7.1 (включая пробу ВК-);
в) в пробирку положительный контроль ПЦР (К+) 10 мкл ПКО Cryptosporidium.
6. Проведение реакции ПЦР РВ с флуоресцентной детекцией
Поместить подготовленные для проведения ПЦР пробирки в ячейки Параметры температурно-временного режима амплификации на приборе «Rotor-Gene Q указаны таблице 3.

Поместить подготовленные для проведения ПЦР пробирки в ячейки амплификатора.
7. Интерпретация результатов анализа
Полученные данные - кривые накопления флуоресцентного сигнала анализируются с помощью программного обеспечения используемого прибора для проведения ПЦР в режиме «реального времени» в соответствии с инструкцией производителя к прибору и в соответствии с Приложениями 1, 2 и 3 к Инструкции.
Учет результатов ПЦР-анализа проводится по наличию или отсутствию пересечения кривой флуоресценции с установленной на соответствующем уровне пороговой линией (что соответствует наличию или отсутствию значения порогового цикла «Ct» для исследуемого образца).
Результат считается достоверным в случае корректного прохождения положительных и отрицательных контролей амплификации и экстракции ДНК в соответствии с таблицей 4.
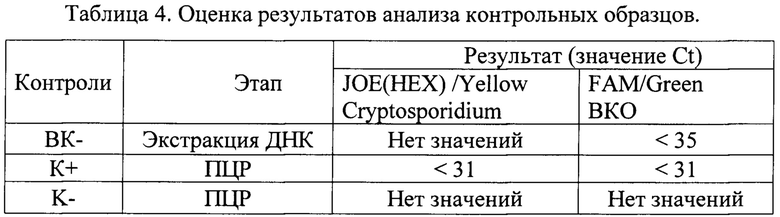
Появление любого значения Ct в таблице результатов для отрицательного контроля этапа экстракции ВК- (на канале JOE(HEX)/Yellow) и для отрицательного контроля этапа ПЦР К- (на любом из каналов) свидетельствует о наличии контаминации реактивов или образцов. В этом случае результаты анализа для всех проб считаются недействительными. Требуется повторить анализ всех проб, а также предпринять меры по выявлению и ликвидации источника контаминации.
Образцы, для которых по каналу FAM/Green значение Ct отсутствует или превышает 35 цикл (и при этом по каналу JOE(HEX)/Yellow отсутствует значение Ct) требуют повторного проведения исследования с этапа ПЦР. Задержка в значениях пороговых циклов для исследуемых образцов на канале FAM/Green указывает на присутствие ингибиторов в пробе(ах) или на ошибки при экстракции ДНК или при постановке реакции. Требуется провести исследование, начиная с этапа экстракции ДНК.
Образец считается положительным (ДНК возбудителя криптоспоридиоза (Cryptosporidium spp.) присутствует) если наблюдается экспоненциальный рост сигнала на канале JOE(HEX)/Yellow, при этом значения Ct контрольных образцов находятся в пределах нормы (табл. 4). Если для исследуемого образца по каналу JOE(HEX)/Yellow значение Ct определяется позднее 35 цикла при корректном прохождении положительных и отрицательных контролей - он считается спорным и исследуется повторно с этапа выделения ДНК. Если при повторной постановке наблюдается схожий результат (Ct на канале JOE(HEX)/Yellow более 35), требуется повторное взятие материала от того же животного для проведения ПНР-исследования и (или) использование альтернативных методов диагностики. В случае получения значения Ct на канале JOE(HEX)/Yellow менее 35 при исследовании повторно взятого от животного материала - образец считать положительным.
Образец считается отрицательным (ДНК возбудителя криптоспоридиоза (Cryptosporidium spp.), отсутствует) если не наблюдается рост сигнала флуоресценции на канале JOE(HEX)/Yellow, при этом значения Ct контрольных образцов находятся в пределах нормы (см. Табл. 4).
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ выявления ДНК возбудителя криптоспоридиоза (Cryptosporidiosis) в биологическом материале животных и кормах с помощью полимеразной цепной реакции в режиме реального времени | 2021 |
|
RU2782427C1 |
| Способ обнаружения ДНК генома возбудителя бордетеллеза (Bordetella bronchiseptica) у сельскохозяйственных животных | 2018 |
|
RU2703405C1 |
| Способ выявления ДНК хламидий у сельскохозяйственных животных и птиц | 2018 |
|
RU2700381C1 |
| Способ выявления ДНК провируса лейкоза крупного рогатого скота (Bovine leukosis virus, BLV) | 2018 |
|
RU2700245C1 |
| Способ выявления генома возбудителя бруцеллезной инфекции (Brucella spp.) у сельскохозяйственных животных | 2018 |
|
RU2703400C1 |
| Тест-система для обнаружения генома возбудителя ДНК Bordetella bronchiseptica инфекции у сельскохозяйственных животных | 2018 |
|
RU2700477C1 |
| Способ выявления ДНК сальмонелл (Salmonella spp.) в биологическом материале животных, продуктах питания и кормах животного и растительного происхождения | 2018 |
|
RU2700476C1 |
| Тест-система для выявления ДНК хламидий у сельскохозяйственных животных и птиц | 2018 |
|
RU2701332C1 |
| Тест-система для выявления ДНК возбудителя моракселлеза KPC (Moraxella bovis) в биологическом материале животных и кормах с помощью полимеразной цепной реакции в режиме реального времени | 2023 |
|
RU2819044C1 |
| Способ выявления ДНК возбудителя лептоспироза (Leptospira spp.) у сельскохозяйственных животных | 2018 |
|
RU2700478C1 |
Изобретение относится к ветеринарной микробиологии, в частности к лабораторной диагностике возбудителей инфекционных заболеваний, а именно к средствам диагностики инфекции у животных с помощью полимеразной цепной реакции. Предложена тест-система для выявления ДНК возбудителя криптоспоридиоза (Cryptosporidiosis) в биологическом материале животных, в воде из водоемов и кормах с помощью полимеразной цепной реакции в режиме реального времени. Тест-система включает буфер для проведения полимеразной цепной реакции, смесь для ее проведения, состоящую из дезоксинуклеозидтрифосфатов, олигонуклеотидных праймеров и флуоресцентных зондов, специфичных для участка генома животного и для внутреннего контрольного образца; смесь ферментов из ДНК полимеразы с антителами, ингибирующих активность фермента, TAQ POLYMERASE, внутренний контрольный образец в виде суспензии бактериофага Т4 с концентрацией 5×103 фаговых частиц на 1 мкл, отрицательный контрольный образец, представляющий собой смесь рекомбинантных плазмидных ДНК, содержащих фрагмент генома животного и фрагмент генома бактериофага Т4; для положительного контрольного образца используют фрагмент генома возбудителя криптоспоридиоза с конкретной нуклеотидной последовательностью; для ДНК возбудителя криптоспоридиоза используют флуоресцентный краситель - JOE(HEX)/Yellow. Тест-система
расширяет функциональные возможности и повышает точность при выявлении возбудителя криптоспоридиоза. 4 табл., 1 пр.
Тест-система для выявления ДНК возбудителя криптоспоридиоза (Cryptosporidiosis) в биологическом материале животных, в воде из водоемов и кормах с помощью полимеразной цепной реакции в режиме реального времени, включающая буфер для проведения полимеразной цепной реакции, смесь для ее проведения, состоящую из дезоксинуклеозидтрифосфатов, олигонуклеотидных праймеров и флуоресцентных зондов, специфичных для участка генома животного и для внутреннего контрольного образца; смесь ферментов из ДНК полимеразы с антителами, ингибирующих активность фермента, TAQ POLYMERASE, внутренний контрольный образец в виде суспензии бактериофага Т4 с концентрацией 5×103 фаговых частиц на 1 мкл, отрицательный контрольный образец, представляющий собой смесь рекомбинантных плазмидных ДНК, содержащих фрагмент генома животного и фрагмент генома бактериофага Т4 с нуклеотидной последовательностью:
T4F: 5`-TACATATAAATCACGCAAAGC-3` - прямой праймер
T4R: 5`-TAGTATGGCTAATCTTATTGG-3` - обратный праймер
Т4Р: СУ5-5`-ACATTGGCACTGACCGAGTTC-3`-BHQ1 - зонд, взятых в объемном соотношении 1:1, отличающаяся тем, что для положительного контрольного образца используют фрагмент генома возбудителя криптоспоридиоза (Cryptosporidiosis) со следующей нуклеотидной последовательностью:
Crypto-F: 5`-AAGGGTTGTATTTATTAGATAA-3`
Crypto-R: 5`-GTCAGAAACTTGAATGATA-3`
Crypto-Z: НЕХ-5`-TCCGTAAAGTTATTATGAGTCACCAAT-3`-BHQ1,
при этом для ДНК возбудителя криптоспоридиоза (Cryptosporidiosis) используют флуоресцентный краситель - JOE(HEX)/Yellow.
| Тест-система для выявления ДНК вируса нодулярного дерматита (LSDV) в биологическом материале животных с помощью полимеразной цепной реакции в режиме реального времени | 2019 |
|
RU2726242C1 |
| Тест-система для выявления ДНК провируса лейкоза крупного рогатого скота (Bovine leukosis virus, BLV) | 2018 |
|
RU2700450C1 |
| CN 106435008 A, 22.02.2017. | |||
Авторы
Даты
2022-12-07—Публикация
2021-12-24—Подача