Область техники
[1] Настоящее изобретение относится к сигнальной последовательности для экспрессии белка CRM197 в Е. coli и секреции белка CRM197 в периплазму и ее применению и, более конкретно, к сигнальной последовательности для экспрессии белка CRM197, нуклеиновой кислоте, кодирующей указанную сигнальную последовательность, конструкции нуклеиновой кислоты или вектору экспрессии, содержащему нуклеиновую кислоту и ген белка CRM197, рекомбинантному микроорганизму, в который введена конструкция нуклеиновой кислоты или вектор экспрессии, и способу получения белка CRM197, включающему культивирование рекомбинантного микроорганизма.
[2]
Уровень техники
[3] Дифтерийный токсин (DT) представляет собой белковый экзотоксин, синтезируемый и секретируемый патогенным штаммом Corynebacterium diphtheriae. Дифтерийный токсин представляет собой АДФ-рибозилирующий фермент, который секретируется в виде профермента, состоящего из 535 остатков и разделенного на два фрагмента (фрагменты А и В) обработкой трипсиноподобной протеазой. Фрагмент А представляет собой каталитически активную область и представляет собой НАД-зависимую АДФ-рибозилтрансферазу, которая специфически нацелена на фактор синтеза белка EF-2, тем самым инактивируя EF-2 и прерывая синтез белка в клетках.
[4] CRM197 был обнаружен путем выделения различных нетоксичных форм и частично токсичных иммунологически перекрестно-реактивных форм (CRM или перекрестно-реактивные вещества) дифтерийного токсина (Uchida et al., Journal of Biological Chemistry 248, 3845-3850, 1973). Предпочтительно CRM может иметь заданный размер и состав, включающий все или часть DT.
[5] CRM197 представляет собой ферментативно крайне неактивную и нетоксичную форму дифтерийного токсина, которая содержит одну аминокислотную замену (G52E). Эта мутация придает внутреннюю гибкость петле активного сайта, расположенной перед сайтом связывания НАД, тем самым снижая аффинность связывания CRM197 с НАД и устраняя токсичность DT (Malito et al., Proc. Natl. Acad. Sci. USA 109 (14): 5229-342012). CRM197, как и DT, имеет две дисульфидные связи. Одна дисульфидная связь связывает Cys186 с Cys201, тем самым связывая фрагмент А с фрагментом В. Другая дисульфидная связь связывает Cys461 с Cys471 во фрагменте В. DT и CRM197 обладают нуклеазной активностью, приобретенной от фрагмента A (Bruce et al., Proc. Natl. Acad. Sci. USA 87, 2995-8, 1990).
[6] Ряд антигенов обладает низкой иммуногенностью, особенно у младенцев и детей младшего возраста, если только они химически не связаны с белками и, таким образом, образуются в виде конъюгатов или конъюгированных вакцин. В этих конъюгированных вакцинах белковый компонент также называют «белком-носителем». CRM197 обычно используют в качестве белка-носителя в конъюгации белок-углевод и конъюгации гаптен-белок. CRM197, как белок-носитель, имеет несколько преимуществ по сравнению с дифтерийным токсоидом, а также другими токсоидными белками (Shinefield Vaccine, 28: 4335, 2010).
[7] Способы получения дифтерийного токсина (DT) хорошо известны в данной области техники. Например, DT можно получить путем очистки токсина из культуры Corynebacterium diphtheriae с последующей химической детоксикацией или путем очистки рекомбинантного или генетически детоксицированного аналога токсина.
[8] Обилие белков сделало невозможным массовое производство дифтерийных токсинов, таких как CRM197, для использования в вакцинах. Эту проблему ранее решали путем экспрессии CRM197 в Е. coli (Bishai, et al., J. Bacteriol. 169: 5140-5151), и Bishai et al. сообщили о рекомбинантном слитом белке, содержащем токсин (включая сигнальную последовательность токсина), приводящем к продукции деградированных белков.
[9] По сравнению с цитоплазматической продукцией, продукция бактериальных токсинов в периплазме характеризуется тем, что i) белок продуцируется в зрелой форме после расщепления сигнального пептида, или ii) периплазма Е. coli является окислительной средой, что позволяет образовывать дисульфидные связи, которые могут содействовать продукции растворимых, правильно свернутых белков, iii) периплазма Е. coli содержит меньше протеазы, чем цитоплазма, что помогает избежать протеолитического расщепления экспрессируемых белков, и iv) периплазма также содержит меньшее количество белков, что позволяет получать рекомбинантный белок более высокой чистоты.
[10] В общем, присутствие сигнальных последовательностей на белках облегчает транспортировку (прокариотические хозяева) или секрецию (эукариотические хозяева) белков в периплазму. У прокариотических хозяев сигнальные последовательности координируют новообразованные белки с периплазмой через внутреннюю мембрану, а затем расщепляются. То есть важен поиск сигнальных последовательностей, способных более эффективно массово продуцировать коммерчески необходимые белки, и необходимо разрабатывать рекомбинантные микроорганизмы.
[11]
[12] Соответственно, в результате значительных усилий по разработке способа получения белка CRM197 эффективным и экономичным способом, авторы настоящего изобретения выбрали конкретные сигнальные последовательности, сконструировали нуклеотидные последовательности, комбинируя кодоновый контекст со вторичной структурой, для того, чтобы оптимизировать трансляцию в Е. coli, оптимизировать экспрессию в Е. coli нуклеотидных последовательностей CRM197, кодирующих белок CRM197, и обнаружено, что CRM197 эффективно экспрессируется в Е. coli и эффективно секретируется в периплазму без изменения рН при их использовании, тем самым завершая настоящее изобретение.
[13]
[14] Информация, раскрытая в этом разделе «Уровень техники», предоставлена только для лучшего понимания предыстории настоящего изобретения, и поэтому она может не включать информацию, формирующую предшествующий уровень техники, которая уже очевидна для специалистов в данной области техники.
[15]
Краткое описание сущности изобретения
[16] Одной из целей настоящего изобретения является обеспечение сигнальной последовательности для экспрессии белка CRM197, имеющего специфическую последовательность, нуклеиновой кислоты, кодирующей сигнальную последовательность, и способа получения белка CRM197 с использованием нуклеиновой кислоты для максимизации экспрессии белка CRM197 при минимизации токсичности белка CRM197 по отношению к Е. coli за счет секреции белка CRM197 в периплазму Е. coli.
[17]
[18] Для достижения целей настоящее изобретение обеспечивает сигнальную последовательность для экспрессии белка CRM197, представленную любой из аминокислотных последовательностей SEQ ID NO: 13 - SEQ ID NO: 21.
[19] Настоящее изобретение также относится к нуклеиновой кислоте, кодирующей сигнальную последовательность для экспрессии белка CRM197.
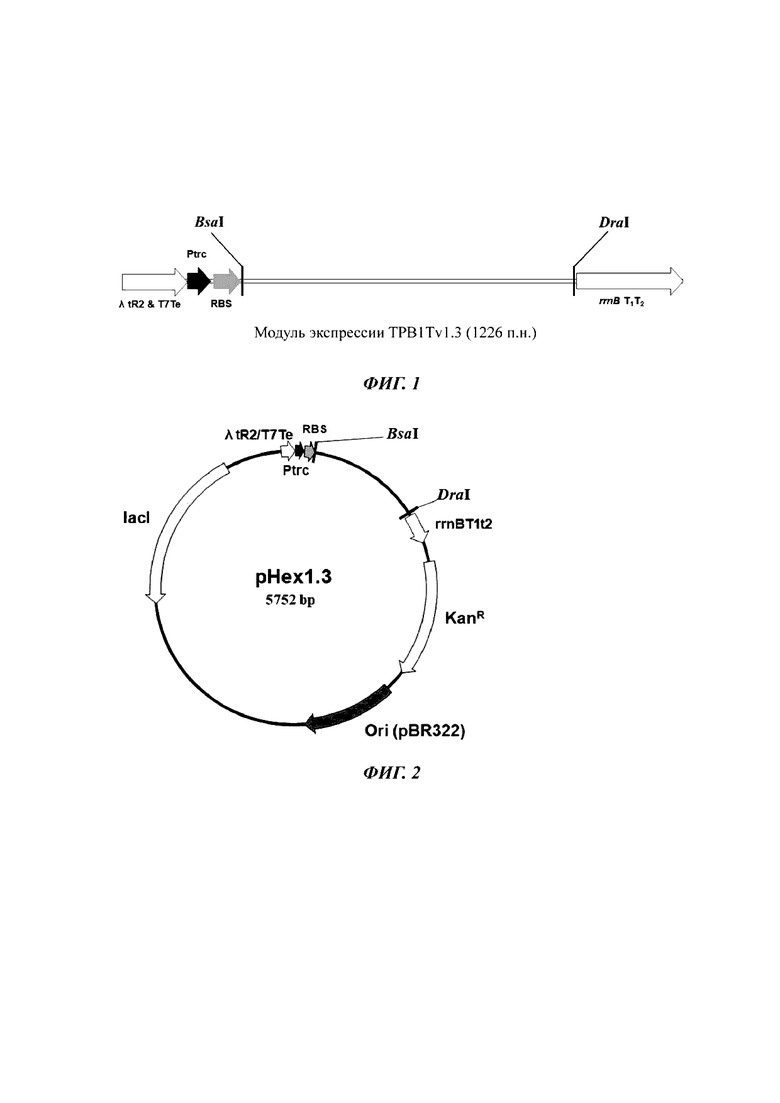
[20] Настоящее изобретение также относится к конструкции нуклеиновой кислоты или вектору экспрессии, содержащему нуклеиновую кислоту и ген белка CRM197, рекомбинантному микроорганизму, в который введена конструкция нуклеиновой кислоты или вектор экспрессии, и способу получения белка CRM197, включающему культивирование рекомбинантного микроорганизма.
[21]
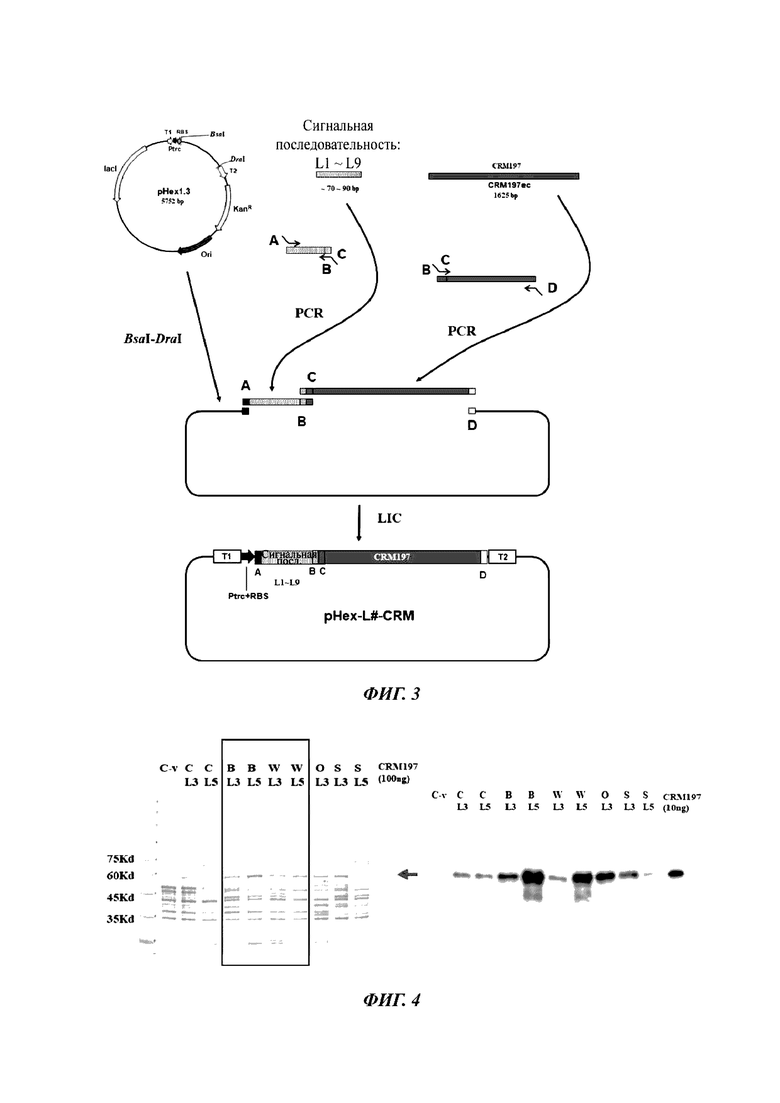
Краткое описание графических материалов
[22] На ФИГ. 1 представлена схематическая диаграмма, иллюстрирующая модуль экспрессии TPB1TV1.3, где Ptrc обозначает промотор trc, RBS обозначает богатый A/U энхансер + SD, λtR2 и Т7Те, a rrnB T1T2 обозначает терминаторы транскрипции, a Bsal и Dral обозначают сайты ферментов рестрикции, используемые для клонирования.
[23] ФИГ. 2 иллюстрирует экспрессионную плазмиду E. coli рНех1.3, где Ori (pBR322) обозначает точку начала репликации pBR322, KanR обозначает маркер канамицина, lacI обозначает ген lacI, а другие символы являются такими же, как на ФИГ. 1.
[24] На ФИГ. 3 представлена схематическая диаграмма, иллюстрирующая получение экспрессионной плазмиды CRM197, где Т1 обозначает терминатор транскрипции λtR2 и Т7Те, Т2 обозначает терминатор транскрипции rrnB Т1Т2, стрелки обозначают праймеры для ПЦР, буквенные символы А, В, С и D обозначают гомологичные области для LIC, а остальные символы являются такими же, как на ФИГ. 1.
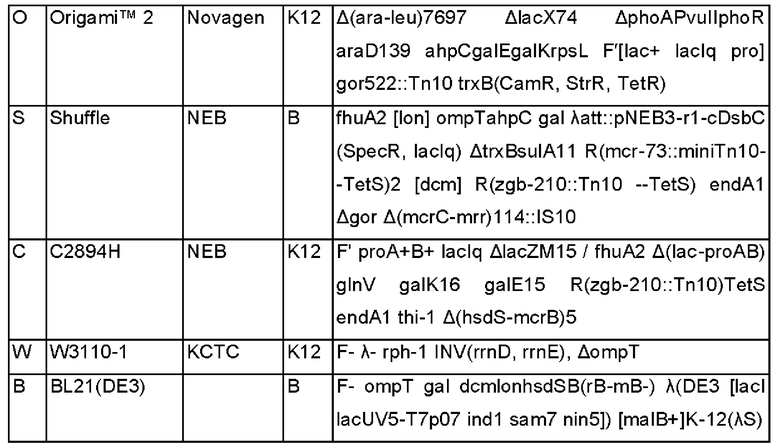
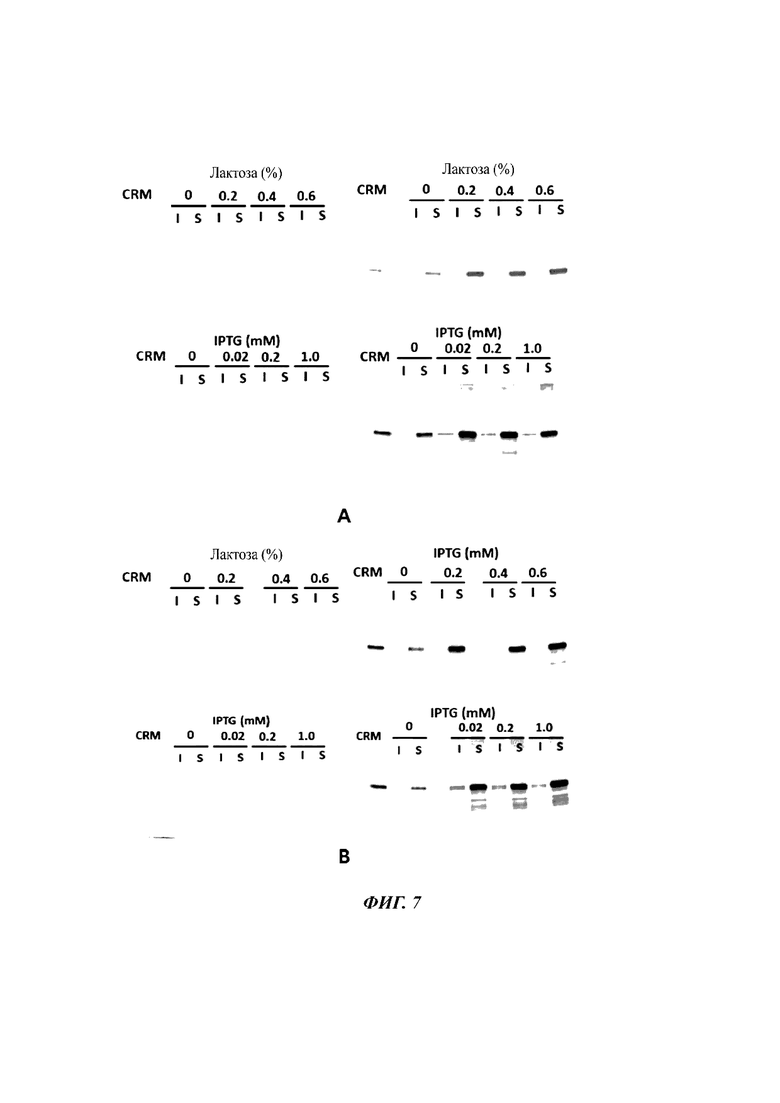
[25] ФИГ. 4 иллюстрирует поведение экспрессии слияния CRM 197 с L3 и L5 в различных штаммах Е. coli, а также результаты окрашивания Кумасси (слева) и Вестерн-блоттинга (справа) после SDS-PAGE всех клеток Е. coli, где С обозначает С2894Н, В обозначает BL21 (DE3), W обозначает W3110-1, О обозначает Origami™ 2, S обозначает «перетасовку», C-v представляет собой С2984Н, содержащую рНех1.3, используемую в качестве отрицательного контроля, L3 и L5 представляют собой слитые сигнальные последовательности, a CRM197 представляет собой эталонный CRM197, и клетки загружают в каждую лунку при плотности, соответствующей OD600, равной 0,025, для окрашивания Кумасси и при плотности, соответствующей OD600, равной 0,0005, для Вестерн-блоттинга.
[26] ФИГ. 5 иллюстрирует влияние типа и концентрации индуктора на экспрессию CRM197, индуцированную L5, и иллюстрирует случаи окрашивания Кумасси (слева) и Вестерн-блоттинга (справа) после SDS-PAGE, где количества загруженных клеток представляют собой такие же, как на ФИГ. 4, I обозначает нерастворимую фракцию, S обозначает растворимую фракцию, (А) показывает культуру при 25°С, а (В) показывает культуру при 30°С.
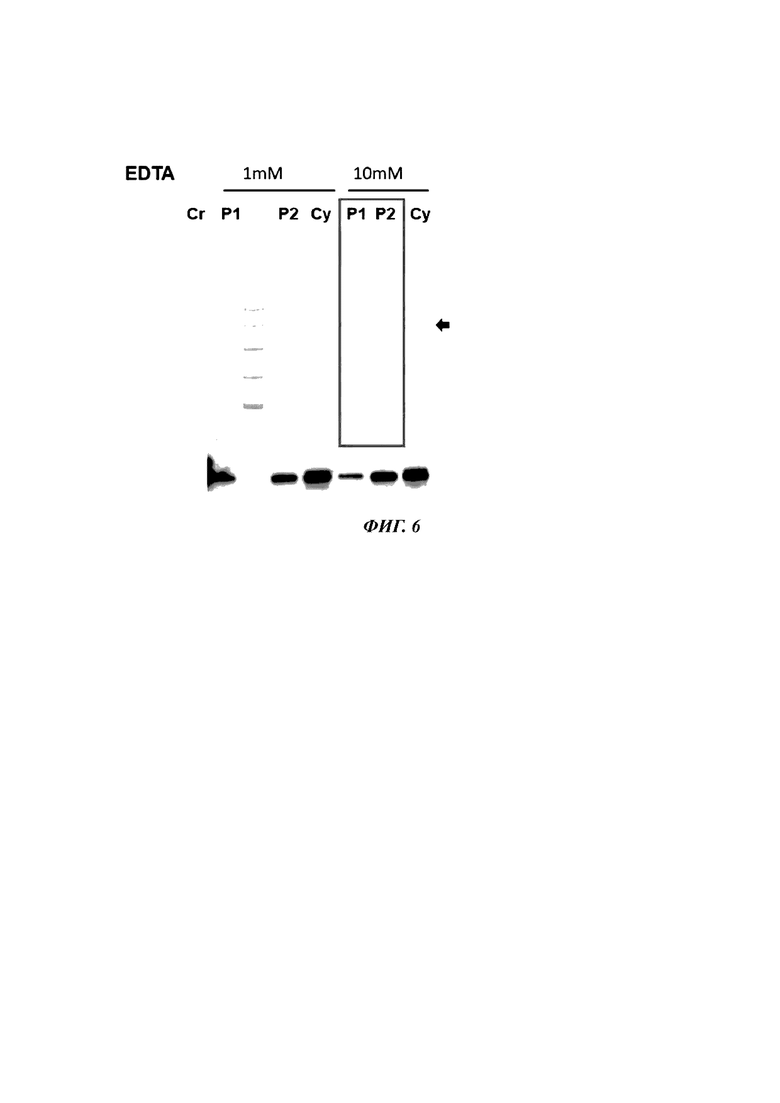
[27] На ФИГ. 6 показано положение белка CRM197, индуцированного слиянием с L5, где вверху показано окрашивание Кумасси после SDS-PAGE, внизу показан Вестерн-блоттинг, Cr представляет собой эталонный CRM197, стрелка показывает положение зрелого CRM197, Р1 обозначает супернатант, полученный после обработки буфером для индукции плазматической мембраны, Р2 обозначает периплазматическую фракцию, а Су обозначает цитоплазматическую фракцию.
[28] ФИГ. 7 иллюстрирует влияние типа и концентрации индуктора на экспрессию CRM197, индуцированную L3, где (А) показывает культуру при 25°С, а (В) показывает культуру при 30°С.
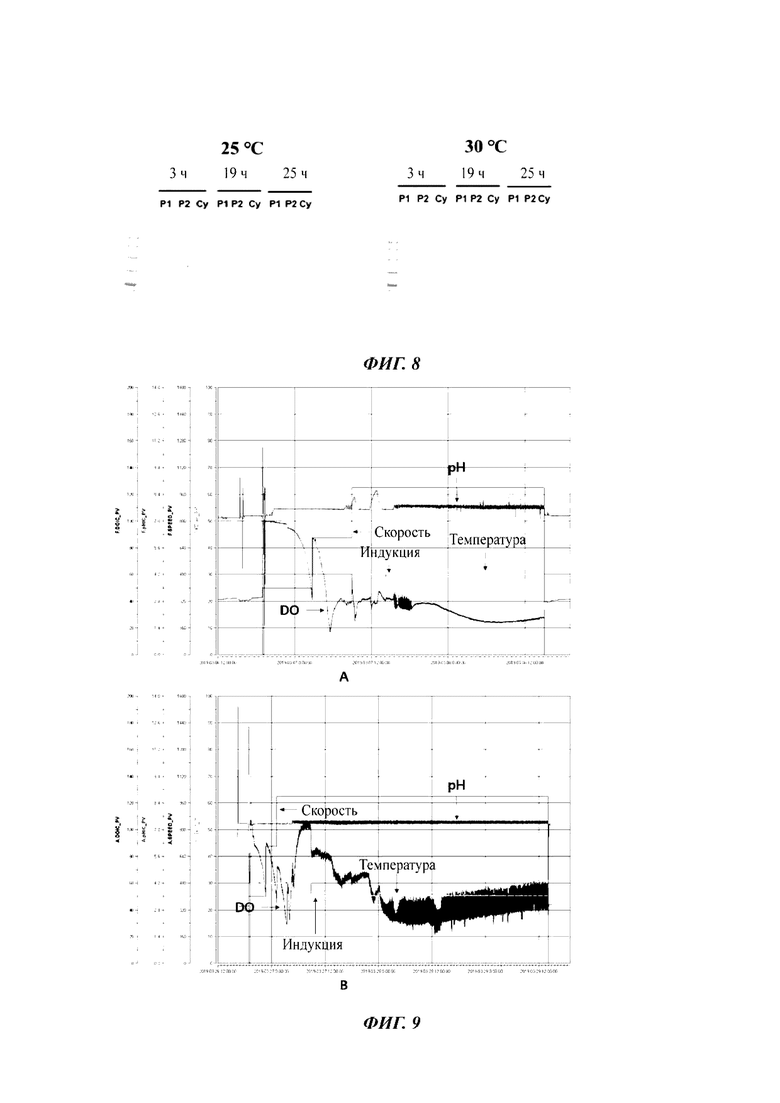
[2 9] На ФИГ. 8 показано положение белка CRM197, индуцированное слиянием с L3.
[30] На ФИГ. 9 показаны изменения рН штамма L3, температуры, скорости лопастной мешалки и растворенного кислорода (DO), а также время добавления индуктора экспрессии, где (А) показывает случай, когда температуру поддерживают на уровне 30°С, а (В) показывает случай, когда перед индукцией экспрессии температуру снижают до 25°С.
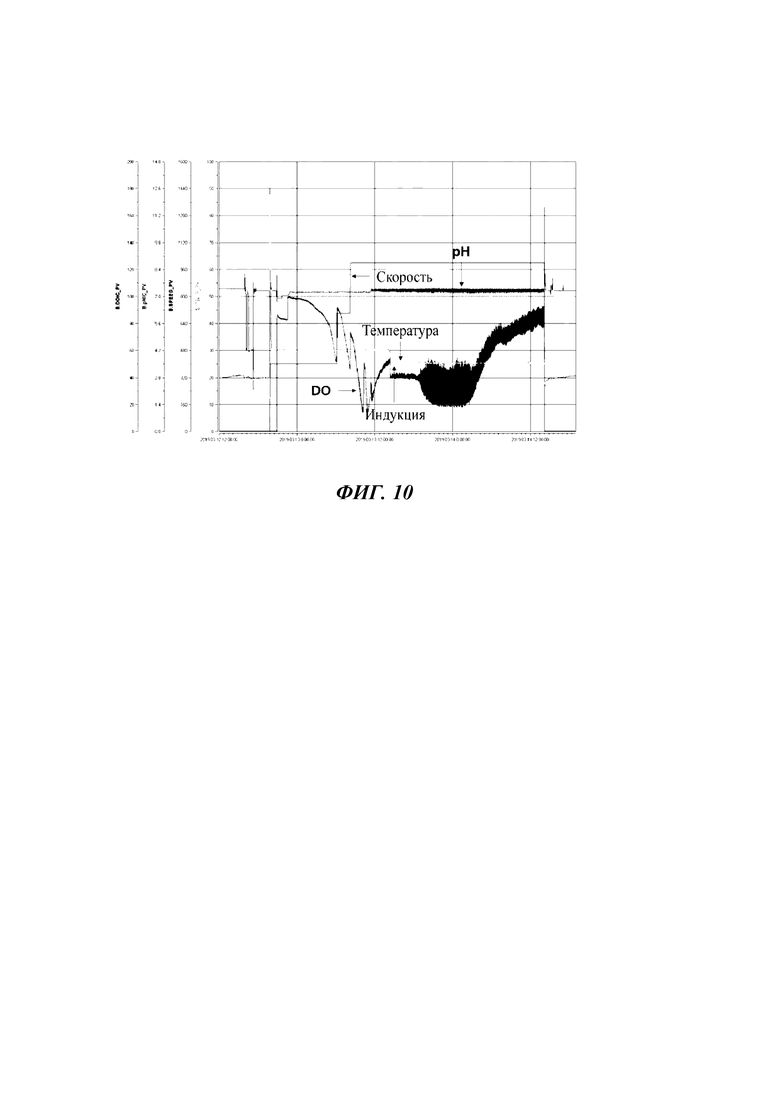
[31] На ФИГ. 10 показаны изменения рН штамма L5, температуры, скорости лопастной мешалки и растворенного кислорода (DO), а также время добавления индуктора экспрессии.
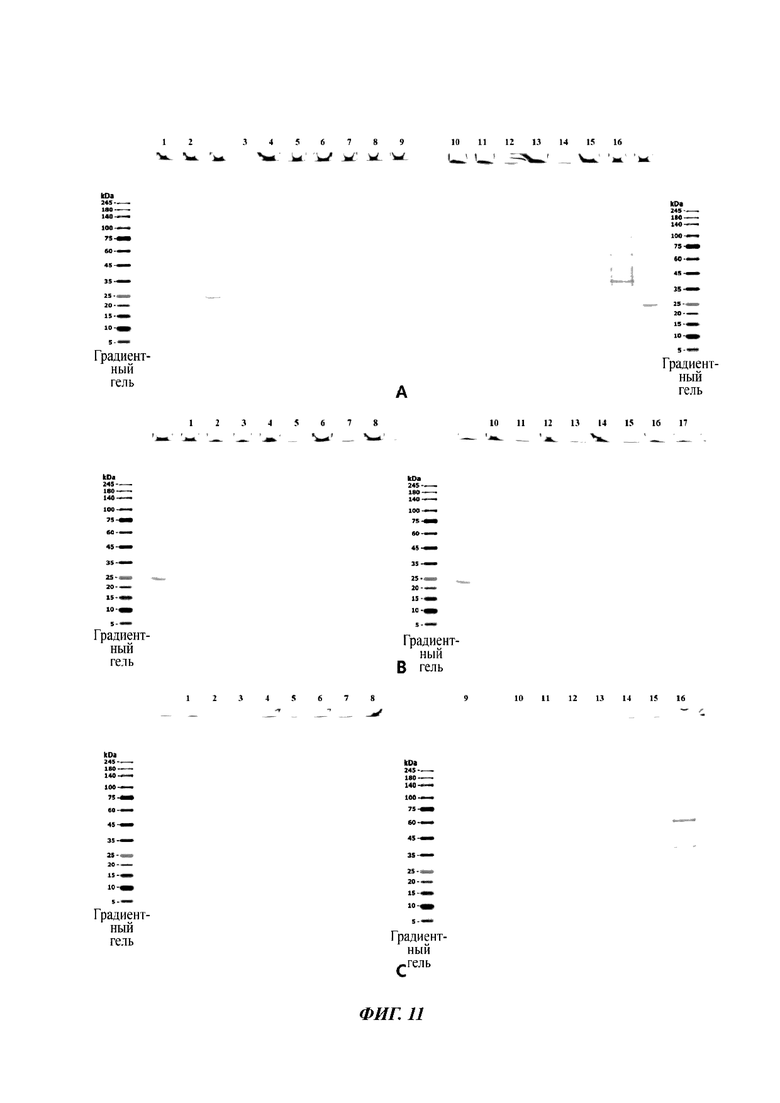
[32] На ФИГ. 11 показано изменение экспрессии белка CRM197 до и после добавления индуктора экспрессии во время культивирования, где (А) показывает экспрессию штамма L3, индуцированную при 30°С, (В) показывает экспрессию штамма L3, индуцированную при 25°С, (С) показывает экспрессию штамма L5, индуцированную при 25°С. 200 нг эталонного CRM197 загружали в линии 3 и 10 в (А), линии 4 и 10 в (В) и линии 3 и 9 в (С). Линии 1 и 2 в (А), линии 1, 2 и 3 в (В) и линии 1 и 2 в (С) представляют собой растворы клеточных культур перед добавлением индуктора экспрессии, а линия 16 в (А) и линия 16 в (В) представляют собой супернатанты после окончания культивирования, последующие линии представляют собой растворы клеточных культур, отбираемые каждые 2 часа после добавления индуктора. Величина загрузки является такой же, как на ФИГ. 4.
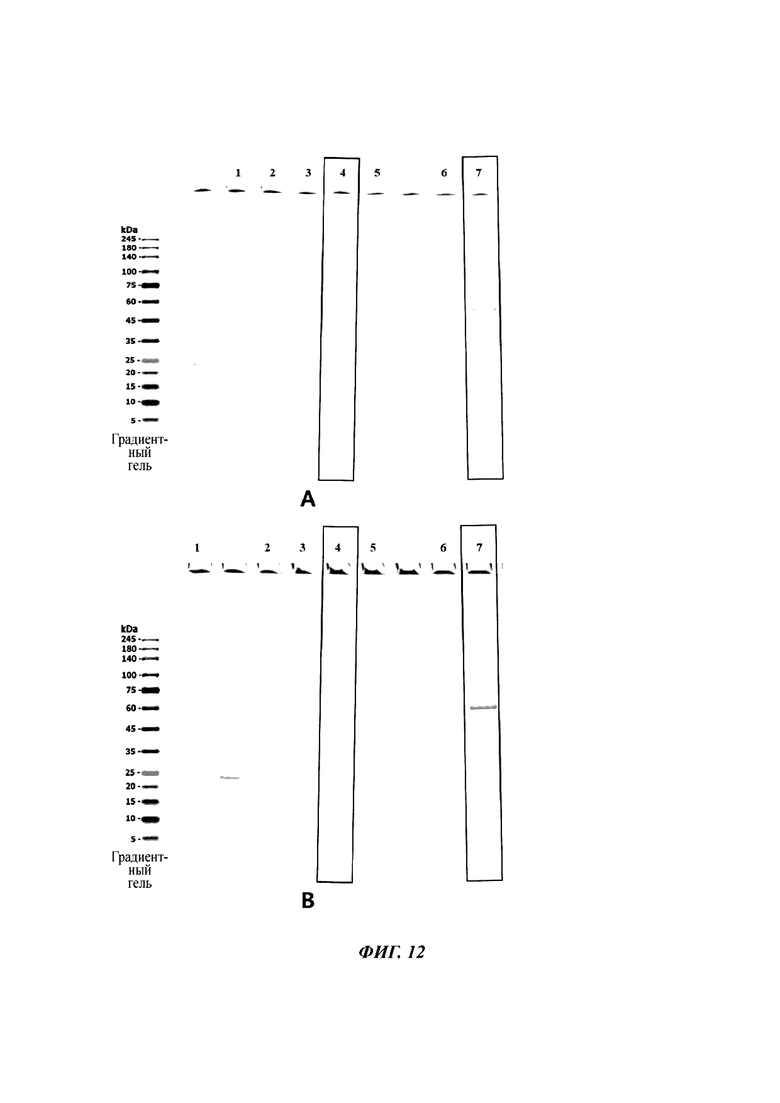
[33] ФИГ. 12 иллюстрирует характер экспрессии и разделения белка CRM197 в штаммах L3/L5, где (А) иллюстрирует поведение белка при разделении в культуре с использованием L3, (В) иллюстрирует поведение белка при разделении в культуре с использованием L5, в линию 1 загружали 200 нг эталонного CRM197, линия 2 представляет собой общий раствор клеточной культуры после завершения культивирования, линия 3 представляет собой супернатант после обработки буфером для индукции плазматической мембраны, линии 4 и 7 представляют собой периплазматические фракции (линия 7 содержит в 4 раза больше белка, чем линия 4), а линии 5 и 6 представляют собой цитоплазматические фракции.
[34] ФИГ. 13 иллюстрирует результат SDS-PAGE образцов, очищенных с помощью DEAE-хроматографии (А) и НА-хроматографии (В). В (А) линия 1 обозначает CRM 197, продуцируемый Corynebacterium, линия 2 обозначает белок, присутствующий в выделенной периплазматической фракции, линия 3 обозначает образец перед загрузкой в DEAE-хроматографию, который дважды концентрируют после ультрафильтрации, линии 4 и 5 используют для обнаружения примесных белков, за исключением CRM197, с использованием проточного режима хроматографии с DEAE и промывными растворами, линия 6 представляет собой элюированный образец, из которого были удалены примеси, линия 7 представляет собой образец, элюированный с использованием высококонцентрированной соли для удаления всех белков, связанных со смолой. НА Elu в (В) представляет собой CRM197, элюированный после удаления белка, не связанного со смолой НА.
[35] ФИГ. 14 иллюстрирует результат анализа SEC-HPLC конечного очищенного CRM197, где чистота составляла 99% или более.
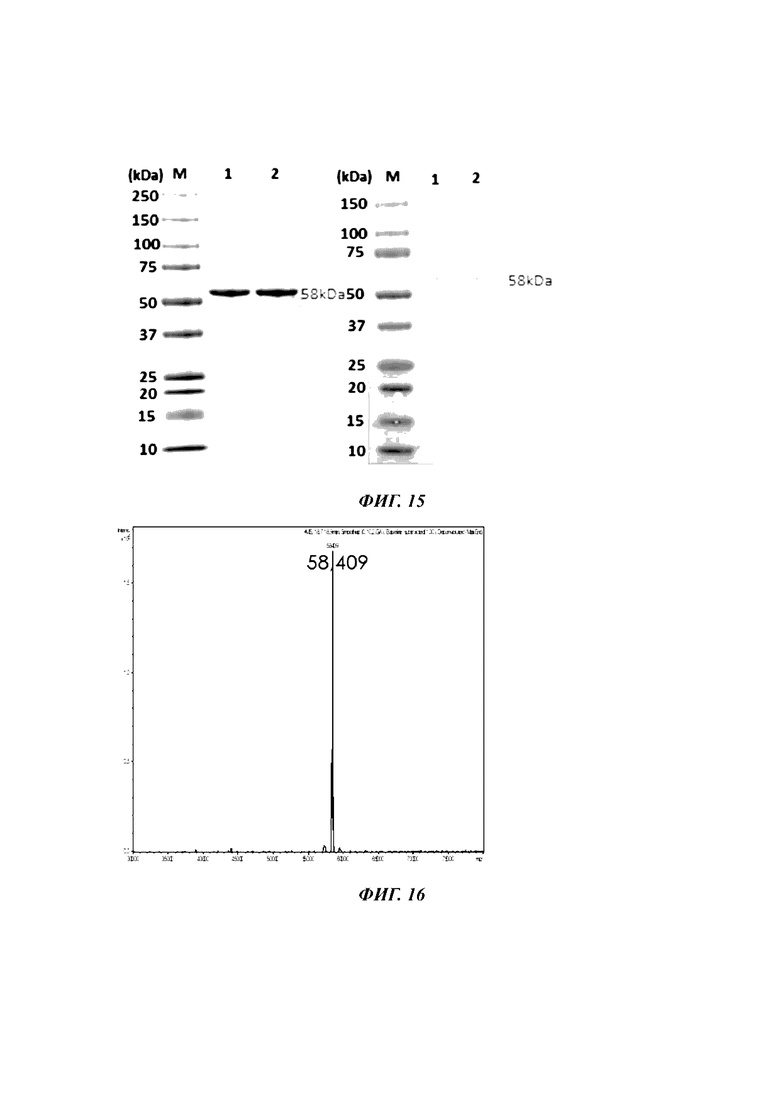
[36] На ФИГ. 15 показаны результаты SDS-PAGE (слева) и Вестерн-блоттинга (справа). Линия 1 представляет собой CRM197, продуцируемый Corynebacterium, а линия 2 представляет собой CRM 197, продуцируемый Е. coli (L3). Оба CRM 197 показали полосы в одном и том же положении.
[37] ФИГ. 16 иллюстрирует результат анализа молекулярной массы интактного белка с использованием ЖХ/МС, где молекулярная масса составляет 58409 Да, что соответствует теоретической молекулярной массе.
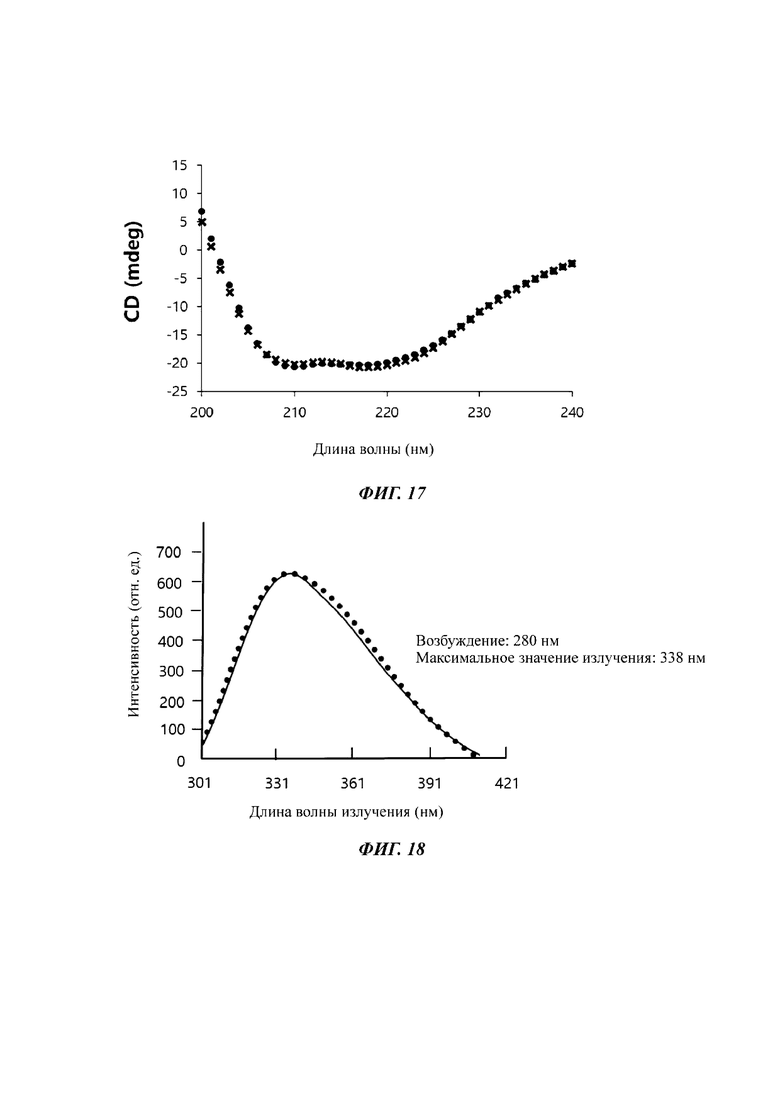
[38] ФИГ. 17 иллюстрирует результат анализа кругового дихроизма (CD) и указывает на отсутствие различий в структуре более высокого порядка между CRM197 (•), продуцируемым Corynebacterium, и CRM197 (Х), продуцируемым Е. coli pHex-L3.
[39] ФИГ. 18 иллюстрирует результат анализа спектра флуоресценции, где CRM197 (•), продуцируемый Corynebacterium, и CRM197 (сплошная линия), продуцируемый Е. coli pHex-L3, имели одинаковую максимальную длину волны излучения 338 нм.
[40]
Подробное описание и предпочтительные варианты осуществления изобретения
[41] Если не указано иное, все технические и научные термины, используемые в данном документе, имеют те же значения, что и подразумевают специалисты в области, к которой относится настоящее изобретение. В общем, используемая в настоящей заявке номенклатура хорошо известна в данной области техники и обычно используется.
[42]
[43] В одном варианте осуществления настоящего изобретения 9 сигнальных последовательностей сливали с белком CRM197 для индукции его экспрессии. Для каждой сигнальной последовательности были сконструированы нуклеотидные последовательности (SEQ ID NO: 4 - SEQ ID NO: 12), оптимизированные для трансляции, с учетом кодонового контекста и вторичной структуры мРНК. Эти конструкции встраивали в экспрессионную плазмиду рНех1.3, и экспрессию CRM197 наблюдали в пяти штаммах Е. coli, чтобы найти оптимальный штамм Е. coli для каждой конструкции. Кроме того, в выбранной Е. coli устанавливали температуру культивирования, а также тип и концентрацию индуктора. В результате трансформации различных штаммов Е. coli конструкциями белок CRM197 мог экспрессироваться в растворимой форме даже в штаммах, у которых не сконструированы гены (trxB, gor), связанные с окислительно-восстановительным потенциалом, и мог секретироваться в периплазме.
[44]
[45] Таким образом, в одном аспекте настоящее изобретение направлено на сигнальную последовательность для экспрессии белка CRM197, представленную любой из аминокислотных последовательностей от SEQ ID NO: 13 по SEQ ID NO: 21.
[46] В другом аспекте настоящее изобретение направлено на нуклеиновую кислоту, кодирующую сигнальную последовательность для экспрессии белка CRM197.
[47] В настоящем изобретении нуклеиновая кислота может быть представлена любой из нуклеотидных последовательностей от SEQ ID NO: 4 по SEQ ID NO: 12, предпочтительно представлена последовательностями нуклеотидов SEQ ID NO: 6 или SEQ ID NO: 8, но не ограничивается этим.
[48] Используемый в контексте данного документа термин «сигнальная последовательность для экспрессии белка CRM197» означает сигнальную последовательность для экспрессии белка CRM197 и секреции белка CRM197 в периплазму.
[49] В одном варианте осуществления настоящего изобретения были выбраны сигнальная последовательность белка, нацеленного на внешнюю мембрану Е. coli, и сигнальная последовательность, полученная из фага М13 (Таблица 3), чтобы секретировать белок CRM197 в периплазму. Нуклеотидные последовательности (от SEQ ID NO: 4 по SEQ ID NO: 12) были сконструированы путем объединения кодонового контекста со вторичной структурой для оптимизации трансляции выбранной сигнальной последовательности в Е. coli.
[50]
[51] В другом аспекте настоящее изобретение направлено на конструкцию нуклеиновой кислоты, содержащую нуклеиновую кислоту, кодирующую сигнальную последовательность для экспрессии белка CRM197, и ген белка CRM197.
[52] В другом аспекте настоящее изобретение относится к вектору экспрессии, содержащему нуклеиновую кислоту, кодирующую сигнальную последовательность для экспрессии белка CRM197, и ген белка CRM197.
[53] В одном варианте осуществления настоящего изобретения последовательность ДНК, кодирующая аминокислотную последовательность белка CRM197 (SEQ ID NO: 3), также была оптимизирована для экспрессии Е. coli (SEQ ID NO: 2). Фрагмент ДНК каждой сконструированной сигнальной последовательности и оптимизированный фрагмент ДНК CRM197 встраивали в плазмиду рНех1.3 (ФИГ. 3).
[54] В настоящем изобретении можно использовать любой ген белка CRM197 без ограничений, если он представляет собой ген, кодирующий белок CRM197. Ген белка CRM197 предпочтительно может быть представлен нуклеотидной последовательностью SEQ ID NO: 2, но не ограничивается этим.
[55] Используемый в настоящей заявке термин «трансформация» означает введение в клетки цепи специфической чужеродной ДНК извне. Микроорганизм-хозяин, содержащий введенную цепь ДНК, называют «трансформированным микроорганизмом». Используемый в настоящей заявке термин «трансформация», означающий введение ДНК в хозяина и придание ДНК реплицируемости внехромосомным фактором или хромосомной интеграцией, указывает на то, что вектор, содержащий полинуклеотид, кодирующий целевой белок, вводят в клетку-хозяина, или полинуклеотид, кодирующий целевой белок, интегрируют в хромосому клетки-хозяина для экспрессии белка, кодируемого полинуклеотидом в клетке-хозяине. Трансформированный полинуклеотид включает как трансформированный полинуклеотид, вставленный в хромосому клетки-хозяина и расположенный внутри нее, так и трансформированный полинуклеотид, расположенный вне хромосомы, при условии, что он может экспрессироваться в клетке-хозяине.
[56] Используемый в настоящей заявке термин «конструкция нуклеиновой кислоты» включает как конструкцию нуклеиновой кислоты, встроенную в хромосому клетки-хозяина и расположенную внутри нее, так и конструкцию нуклеиновой кислоты, расположенную вне хромосомы, при условии, что она может экспрессироваться в хромосоме клетки-хозяина.
[57] Кроме того, используемый в настоящем документе термин «полинуклеотид» используется взаимозаменяемо с термином «нуклеиновая кислота» и включает ДНК и РНК, кодирующие целевой белок. Полинуклеотид может быть введен в любой форме при условии, что он может быть введен в клетку-хозяина и экспрессирован в ней. Например, полинуклеотид может быть введен в клетку-хозяина в виде кассеты экспрессии, которая представляет собой генную конструкцию, содержащую все элементы, необходимые для самоэкспрессии. Кассета экспрессии обычно содержит промотор, сигнал терминации транскрипции, сайт связывания рибосомы и сигнал терминации трансляции, который функционально связан с нуклеиновой кислотой. Кассета экспрессии может содержать вектор экспрессии, обеспечивающий саморепликацию. Полинуклеотид также может быть введен в клетку-хозяина в его нативной форме и функционально связан с последовательностью, необходимой для экспрессии в клетке-хозяине.
[58] Используемый в настоящей заявке термин «вектор» означает продукт ДНК, содержащий последовательность ДНК, функционально связанную с подходящей регуляторной последовательностью, способной экспрессировать ДНК в подходящем хозяине. Векторы могут представлять собой плазмиды, фаговые частицы или простые потенциальные геномные вставки. При трансформации в подходящего хозяина векторы могут реплицироваться или выполнять функции, независимые от геномов хозяина, или некоторые из них могут быть интегрированы с геномами. Плазмиды в настоящее время являются наиболее часто используемой формой вектора. Таким образом, термины «плазмида» и «вектор» используются взаимозаменяемо.
[59] С учетом целей настоящего изобретения использование плазмидного вектора является предпочтительным. Типичные плазмидные векторы, которые можно использовать для достижения задач, включают (а) точку начала репликации для эффективного проведения репликации, чтобы включать несколько или несколько сотен плазмидных векторов в каждой клетке-хозяине, (b) ген устойчивости к антибиотикам для скрининга клеток-хозяев, трансформированных плазмидными векторами и (с) сайт расщепления ферментом рестрикции, в который вставлен фрагмент чужеродной ДНК. Даже если соответствующий сайт расщепления ферментом рестрикции отсутствует, вектор и чужеродная ДНК могут быть легко лигированы с использованием синтетического олигонуклеотидного адаптера или линкера в соответствии с обычным способом.
[60] Кроме того, когда ген выровнен с другой последовательностью нуклеиновой кислоты на основе функциональной взаимосвязи между ними, говорят, что он «функционально связан» с ней. Это могут быть ген(ы) и регуляторная(ые) последовательность(и), связанные таким образом, чтобы обеспечить экспрессию гена, когда подходящая молекула (например, белок-активатор транскрипции) связана с регуляторной(ыми) последовательностью(ями). Например, ДНК препоследовательности или секреторного лидера функционально связана с ДНК полипептида, когда она экспрессируется в виде препротеина, участвующего в секреции полипептида; промотор или энхансер функционально связан с кодирующей последовательностью, когда он влияет на транскрипцию последовательности; сайт связывания рибосомы функционально связан с кодирующей последовательностью, когда он влияет на транскрипцию последовательности; или сайт связывания рибосомы функционально связан с кодирующей последовательностью, когда он расположен для облегчения трансляции.
[61] Как правило, термин «функционально связанный» означает, что связанная последовательность ДНК находится в контакте или что секреторный лидер находится в контакте с ней и присутствует в рамке считывания. Однако энхансер не должен контактировать с ними. Связывание этих последовательностей осуществляется путем лигирования в удобных сайтах рестрикционных ферментов. Когда такого сайта не существует, используют синтетический олигонуклеотидный адаптер или линкер в соответствии с обычным способом.
[62] В настоящем изобретении вектор экспрессии может дополнительно содержать промотор Trc.
[63] В настоящем изобретении вектор экспрессии может представлять собой рНех1.3, но не ограничивается этим.
[64] Известно, что CRM197 является высокотоксичным для Е. coli из-за его нуклеазной активности. Таким образом, экспрессия CRM197 в нежелательных условиях может отрицательно сказаться на росте Е. coli. Экспрессионная плазмида рНех1.3 Е. coli содержит ген Lacl и может подавлять фоновую экспрессию промотора trc, а встроенный в эту плазмиду модуль экспрессии TPB1Tv1.3 (ФИГ. 1) предназначен для подавления экспрессии CRM197, транскрибируемого с промотора, полученного из плазмиды, путем встраивания терминатора транскрипции tR2, полученного из фага λ, и терминатора транскрипции Те, полученного из фага Т7, в вышерасположенную часть экспрессируемого гена CRM197, и терминатора транскрипции rrnB Т1Т2, полученного из Е. coli, в нижерасположенную его часть.
[65]
[66] В другом аспекте настоящее изобретение направлено на рекомбинантный микроорганизм, в который введена конструкция нуклеиновой кислоты или вектор экспрессии.
[67] В настоящем изобретении рекомбинантный микроорганизм может представлять собой Escherichia coli, но не ограничивается этим.
[68] В качестве рекомбинантного микроорганизма обычно используют клетки-хозяева, обладающие высокой эффективностью введения ДНК и высокой эффективностью экспрессии введенной ДНК, все бактерии, дрожжи, плесень и т.д., то есть все микроорганизмы, включая прокариотические и эукариотические клетки, доступны, и в примере по настоящему изобретению использовали Е. coli, но настоящее изобретение не ограничивается ими, и можно использовать любой тип микроорганизмов при условии, что белок CRM197 может быть экспрессирован в достаточной степени.
[69] Следует понимать, что не все векторы функционируют одинаково при экспрессии последовательностей ДНК согласно настоящему изобретению. Точно так же не все хозяева одинаково функционируют для одной и той же системы экспрессии. Однако специалисты в данной области техники смогут сделать соответствующий выбор среди различных векторов, последовательностей, регулирующих экспрессию, и хозяев без чрезмерного бремени экспериментов и без отклонения от объема настоящего изобретения. Например, выбор вектора следует проводить с учетом хозяина, поскольку вектор должен реплицироваться в нем. Также следует учитывать количество репликатов вектора, возможность контролировать количество репликатов вектора и экспрессию других белков, кодируемых соответствующим вектором, таких как экспрессия антибиотических маркеров.
[70] Трансформированный рекомбинантный микроорганизм может быть получен любым известным способом трансформации.
[71] В настоящем изобретении способ встраивания гена в хромосому клеток-хозяев может быть выбран из общеизвестных способов генетических манипуляций, например, способов с использованием ретровирусных векторов, аденовирусных векторов, аденоассоциированных вирусных векторов, векторов вирусов простого герпеса, поксвирусных векторов, лентивирусных векторов или невирусных векторов.
[72] Кроме того, трансформацию можно осуществить путем прямой вставки конструкции нуклеиновой кислоты в хромосому клеток-хозяев в дополнение к использованию векторов экспрессии.
[73] В целом, можно использовать электропорацию, липофекцию, баллистическую доставку, виросомы, липосомы, иммунолипосомы, поликатионы или конъюгаты липид:нуклеиновая кислота, «голую» ДНК, искусственные вирионы, химически стимулированный приток ДНК, осаждение фосфата кальция (CaPO4), осаждение хлорида кальция (CaCl2), микроинъекцию, метод ацетата лития-ДМСО и т.д.
[74] Сонопорация, например, способы с использованием системы Sonitron 2000 (Rich-Mar), также могут быть использованы для доставки нуклеиновых кислот, а другие репрезентативные системы доставки нуклеиновых кислот включают Amaxa Biosystems (Cologne, Germany), Maxcyte, Inc. (Rockville, Maryland) и BTX Molecular System (Holliston, Mass.). Способы липофекции раскрыты в патенте США №5049386, патенте США №4946787 и патенте США №4897355, а реагенты для липофекции являются коммерчески доступными, например, TRANSFECTAM™ и LIPOFECTIN™. Катионные или нейтральные липиды, подходящие для эффективной распознающей рецептор липофекции полинуклеотидов, включают липиды Фельгнера (WO 91/17424 и WO 91/16024), которые могут быть доставлены в клетки посредством трансдукции ех-vivo и в ткани-мишени посредством трансдукции in vivo. Способы получения комплекса липид : нуклеиновая кислота, содержащего целевую липосому, такую как иммунолипидный комплекс, хорошо известны в данной области техники (Crystal, Science., 270: 404-410, 1995; Blaese et al., Cancer Gene Ther., 2: 291-297, 1995; Behr et al., Bioconjugate Chem., 5: 382389, 1994; Remy et al., Bioconjugate Chem., 5: 647-654, 1994; Gao et al., Gene Therapy., 2: 710-722, 1995; Ahmad et al., Cancer Res., 52: 4817-4820, 1992; патент США №4186183; патент США №4217344; патент США №4235871; патент США №4261975; патент США №4485054, патент США №4501728, патент США №4774085, патент США №4837028, патент США №4946787).
[75] В одном варианте осуществления настоящего изобретения в результате трансформации различных штаммов Е. coli полученным плазмидным вектором (Таблица 4), для слияния с L3 CRM197 экспрессировался во всех штаммах, и для слияния с L5 CRM197 экспрессировался в штаммах, за исключением Origami™ 2 (ФИГ. 4). Гибриды L3 и L5 были способны экспрессировать белок CRM197 в растворимой форме даже в штаммах, в которых гены (trxB, gor), связанные с окислительно-восстановительным потенциалом, не были сконструированы, и белок имел те же физико-химические/иммунологические свойства, что и белок, выделенный из родительских штаммов. Кроме того, в случае слияния с L3 белок CRM197 может экспрессироваться в растворимой форме как при 25°С, так и при 30°С (ФИГ. 7 и 12А).
[76] Предыдущий отчет показал, что при культивировании при рН от 6,5 до 6,8 и последующем изменении рН до 7,5 во время индукции секреция CRM197 в периплазму улучшается. Напротив, было обнаружено, что штамм, полученный в соответствии с настоящим изобретением, эффективно секретирует CRM197 в периплазму без изменения рН среды (ФИГ. 12). В случае слияния с L5 в высококонцентрированной культуре без смещения рН продуктивность CRM197 составляла 3,7 г/л, а в периплазму секретировалось 2 г/л и более CRM197.
[77]
[78] В другом аспекте настоящее изобретение относится к способу получения белка CRM197, включающему (а) культивирование рекомбинантного микроорганизма, в который введена конструкция нуклеиновой кислоты или вектор экспрессии, для получения белка CRM197, и (b) выделение полученного белка CRM197.
[79] В настоящем изобретении стадия (b) может включать выделение белка CRM197, секретируемого в периплазму.
[80]
[81] Далее настоящее изобретение будет описано более подробно со ссылкой на примеры. Однако специалистам в данной области техники будет очевидно, что эти примеры представлены только для иллюстрации настоящего изобретения и не должны рассматриваться как ограничивающие объем настоящего изобретения.
[82]
[83] Пример 1: Получение сверхэкспрессионной плазмиды CRM197
[84] Пример 1.1: Конструкция плазмиды рНех1.3
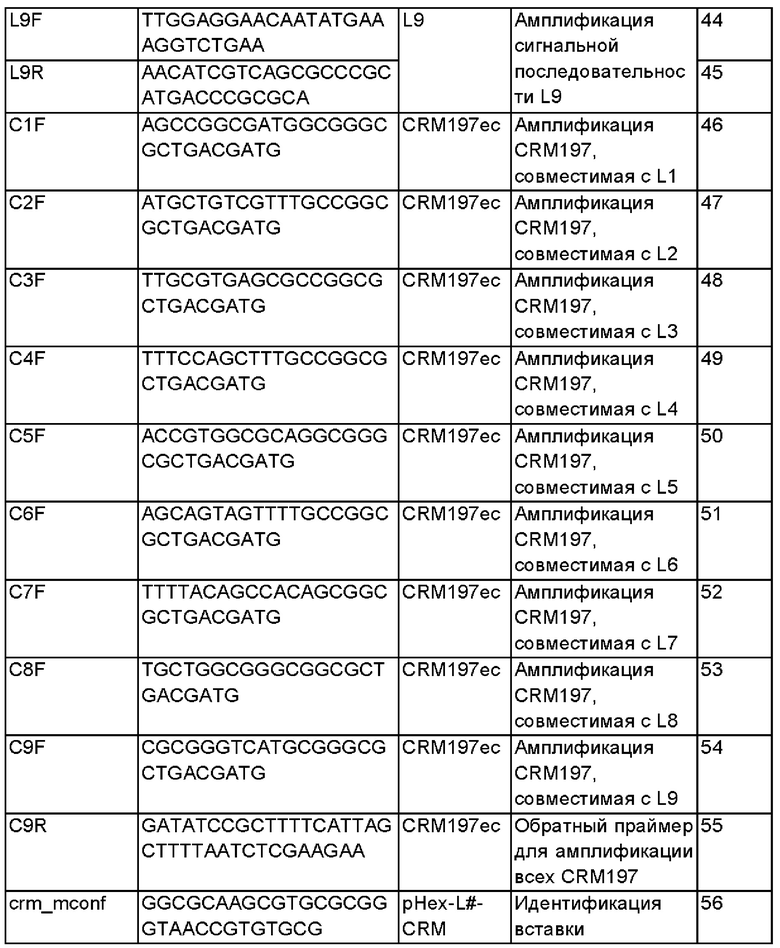
[85] Экспрессионная плазмида Е. coli рНех1.3 была получена следующим образом. После двойного расщепления ptrc99a (Amann et al., Gene. 69, 301-15, 1988) Sspl и Dral фрагмент ДНК размером примерно 3,2 т.п.н. очищали с помощью электрофореза в агарозе. Ген устойчивости к канамицину амплифицировали с помощью ПЦР. Используемая в настоящем документе матрица представляла собой плазмиду pCR2.1, а используемые в настоящем документе праймеры представляли собой KF2 и KR (Таблица 1).
[86]


[88]
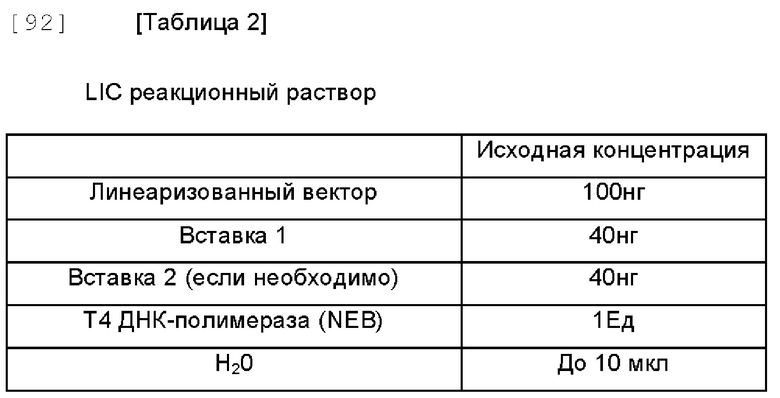
[89] Реакционный раствор для ПЦР готовили с использованием 2,5 мМ каждого dNTP, 10 пмоль каждого праймера, от 200 до 500 нг матричной ДНК, 1,25 Ед ДНК-полимеразы PrimeSTAR HS (Takara Bio Inc., Япония) и 50 мкл реакционного объема, и ПЦР проводили в течение 30 циклов, каждый из которых включал три стадии при 98°С в течение 10 секунд, при 60°С в течение 5 секунд и при 72°С мин/т.п.н. После двойного расщепления фрагмента ДНК размером примерно 0,8 т.п.н., полученного в условиях ПЦР с использованием SamHI и HindIII, фрагмент ДНК дополняли фрагментом Кленова с образованием тупого конца, а затем лигировали с фрагментом ДНК размером 3,2 т.п.н., полученным в предыдущем процессе, используя ДНК-лигазу Т4. Этот реакционный раствор трансформировали в E. coli С2984Н для получения рНех1.1, в котором маркер селекции был заменен с Amp на Km.
[90] Модуль экспрессии TPB1Bv1.3, включающий промоторы, RBS и терминаторы транскрипции (ФИГ. 1), был синтезирован в Bioneer (SEQ ID NO: 1). Процесс загрузки модуля экспрессии TPB1Bv1.3 на рНех1.3 выглядит следующим образом. Фрагмент ДНК (1255 п.н.), полученный с помощью ПЦР-амплификации с использованием модуля экспрессии TPB1Bv1.3 в качестве матрицы и TPB_F и TPB_R в качестве праймеров (Таблица 1), был собран in vitro с фрагментом ДНК (4561 п.н.), полученным с помощью ПЦР-амплификации с использованием рНех1.1. в качестве матрицы и pHex_F и pHex_R в качестве праймеров посредством независимого от лигирования клонирования (LIC; Jeong et al., Appl Environ Microbiol. 78, 5440-3, 2012) в условиях, показанных в Таблице 2, и полученный продукт трансформировали в Е. coli С2984Н для получения рНех1.3 (ФИГ. 2).
[91]
[93]
[94] Условия LIC реакции: Векторы расщепляли рестрикционными ферментами или получали линейные фрагменты ДНК с помощью ПЦР. Вставки готовили с помощью ПЦР. Векторы смешивали со вставками, как показано в таблице выше, а затем обеспечивали реакцию при комнатной температуре в течение 2 минут и 30 секунд.
[95]
[96] Пример 1.2: Конструкция гена CRM197
[97] Нуклеотидная последовательность (CRM197ec) CRM197, оптимально экспрессируемая в Е. coli, была синтезирована в GenScript (SEQ ID NO: 2). Аминокислотная последовательность, кодируемая CRM197ec, представляет собой SEQ ID NO: 3.
[98]
[99] Пример 1.3: Конструирование гена сигнальной последовательности
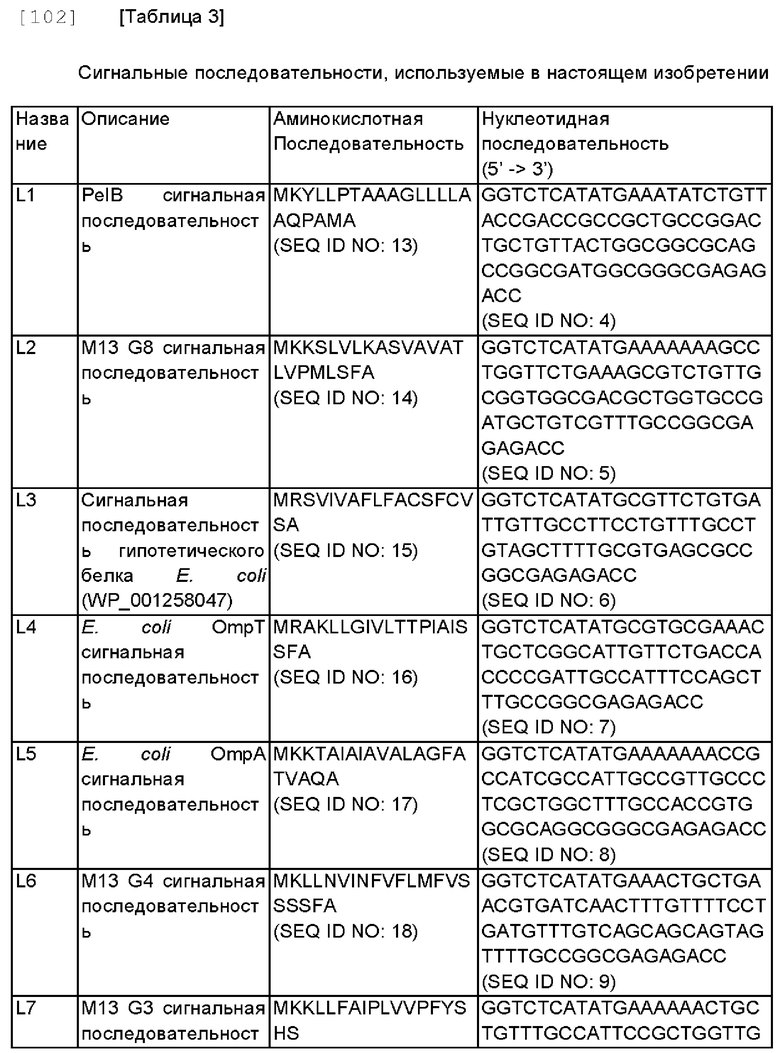
[100] Сигнальные последовательности, используемые для секреции белка CRM197 в периплазму Е. coli, показаны в Таблице 3 ниже (SEQ ID NO: 13-21). Для оптимизации экспрессии в Е. coli синтезировали ДНК с SEQ ID NO: 4-12 с учетом кодонового контекста и вторичной структуры (Таблица 3).
[101]

[103]
[104] Пример 1.4: Конструирование плазмиды для сверхэкспрессии CRM197
[105] Плазмиды для сверхэкспрессии CRM 197 в Е. coli и секреции CRM 197 в периплазму получали, как показано на ФИГ. 3. После двойного расщепления рНех1.3 с помощью BsaI и DraI с помощью электрофореза в агарозе был выделен фрагмент ДНК длиной примерно 5 т.п.н. Сигнальные последовательности L1-L9 амплифицировали с помощью ПЦР с использованием праймеров и матриц, показанных в Таблице 1. Фрагмент ДНК CRM197 для слияния гена CRM197 с каждой сигнальной последовательностью с использованием метода LIC амплифицировали с помощью ПЦР с использованием праймеров и матриц, показанных в Таблице 1. рНех1.3, расщепленный BsaI и DraI, каждый фрагмент сигнальной последовательности и совместимый с ним фрагмент CRM197 собирали in vitro с использованием описанных выше условий LIC, а затем полученный продукт трансформировали в Е. coli С2984Н для получения плазмид pHex-L1-CRM, pHex-L2-CRM, pHex-L3-CRM, pHex-L4-CRM, pHex-L5-CRM, pHex-L6-CRM, pHex-L7-CRM, pHex-L8-CRM и pHex-L9-CRM, содержащих CRM197, слитый с соответствующими сигнальными последовательностями.
[106]
[107] Пример 2: Экспрессия белка CRM197 в Е. coli
[108] pHex-L3-CRM и pHex-L5-CRM были выбраны из плазмид, полученных в Примере 1, с учетом уровня экспрессии и степени клеточного роста. После трансформации pHex-L3-CRM и pHex-L5-CRM в штамм E.coli из Таблицы 4 ниже оценивали экспрессию и положение экспрессии белка CRM197.
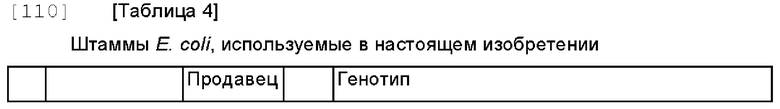
[109]
[111]
[112] Способ культивирования заключается в следующем. Колонию, полученную на твердой среде (10 г/л сойтона, 5 г/л дрожжевого экстракта, 10 г/л NaCl, 15 г/л агара), культивировали при встряхивании в жидкой среде LB, содержащей 100 мМ фосфата калия (рН 7,5), км 50 мкг/мл и 0,2% лактозы, все клетки подвергали SDS-PAGE, а затем анализировали экспрессию CRM197 с использованием окрашивания Кумасси и вестерн-блоттинга (ФИГ. 4). Для слияния с L3 белок CRM197 экспрессировали во всех используемых штаммах, тогда как для слияния L5 рост был невозможен в жидкой среде при трансформации в Origami™ 2. В других штаммах наблюдалась экспрессия белка CRM197.
[113]
[114] Пример 2.1: Экспрессия CRM197 с помощью L5
[115] BL21 (DE3), содержащий pHex-L5-CRM, культивировали в жидкой среде LB (колба с перегородкой 50 мл/500 мл), содержащей 100 мМ фосфата калия (рН 7,5) и 50 мкг/мл km, пока OD600 не достигла 0,4-0,6. Затем в качестве индуктора для индукции экспрессии добавляли 0,2, 0,4 или 0,6% лактозы или 0,02, 0,2 или 2 мМ IPTG (изопропил-β-D-1-тиогалактопианозида). Температура культуры составляла 25°С или 30°С. После культивирования клетки извлекали, суспендировали в 50 мМ фосфате калия (рН 7,0) и затем разрушали ультразвуком. После разрушения проводили центрифугирование для отделения супернатанта (растворимая фракция) от осадка (нерастворимая фракция). После SDS-PAGE каждого образца экспрессию белка CRM197 анализировали с использованием окрашивания Кумасси и вестерн-блоттинга (ФИГ. 5). Когда добавляли индуктор (лактозу, IPTG) и затем проводили культивирование при 25°С, большая часть экспрессированного белка CRM197 присутствовала в растворимой форме и имела ту же молекулярную массу, что и эталонный CRM197, что указывает на то, что экспрессированный белок был зрелым CRM197, из которого была удалена сигнальная последовательность L5 (ФИГ. 5А). С другой стороны, когда в качестве индуктора использовали лактозу и проводили культивирование при 30°С, примерно 50% белка CRM197 было обнаружено в нерастворимой фракции, а когда в качестве индуктора добавляли IPTG, большая часть белка CRM197 была обнаружена в нерастворимой фракции (ФИГ. 5В).
[116] Периплазматическая фракция была извлечена с помощью осмотического шока для определения места, в котором экспрессировался CRM197. Процесс заключается в следующем. BL21 (DE3), содержащий pHex-L5-CRM, культивировали при 25°С и затем центрифугировали для выделения клеток. Клетки ресуспендировали в буфере для индукции плазматической мембраны [30 мМ Tris-HCI (рН 8,0), 20% сахарозы, 1 или 10 мМ ЭДТА, 1 мМ PMSF (фенилметилсульфонилфторид)] до тех пор, пока концентрация клеток не достигала OD600, равной 10, и перемешивали при комнатной температуре от 0,5 до 1 часа. Затем клетки собирали центрифугированием при 4000 × g в течение 15 минут и добавляли такое же количество 30 мМ холодного (4°С или ниже) Tris-HCI (рН 8,0) с последующим перемешиванием при комнатной температуре в течение 0,5-1 часа. Затем клетки центрифугировали при 4000 g в течение 15 минут для получения супернатанта (периплазматическая фракция, Р2). После обработки буфером для индукции плазматической мембраны супернатант (Р1), периплазматическую фракцию (Р2) и цитоплазматическую фракцию проявляли с помощью SDS-PAGE, а затем оценивали экспрессию CRM197 и положение экспрессированного CRM197 с использованием окрашивания Кумасси и вестерн-блоттинга (ФИГ. 5). Было обнаружено, что L5 может успешно секретировать CRM197 в периплазму (ФИГ. 6). ЭДТА в качестве буфера для индукции плазматической мембраны была более эффективной при 10 мМ, чем при 1 мМ.
[117]
[118] Пример 2.2: Экспрессия CRM197 с помощью L3
[119] BL21 (DE3), содержащий pHex-L3-CRM, экспрессировали в тех же условиях, что и в примере 2.1. Было обнаружено, что CRM197, индуцированный L3, в отличие от L5, присутствовал в растворимой форме при любых условиях (ФИГ. 7) и секретировался в периплазму (ФИГ. 8).
[120]
[121] Пример 3: Культура Е. coli BL21 (DE3)
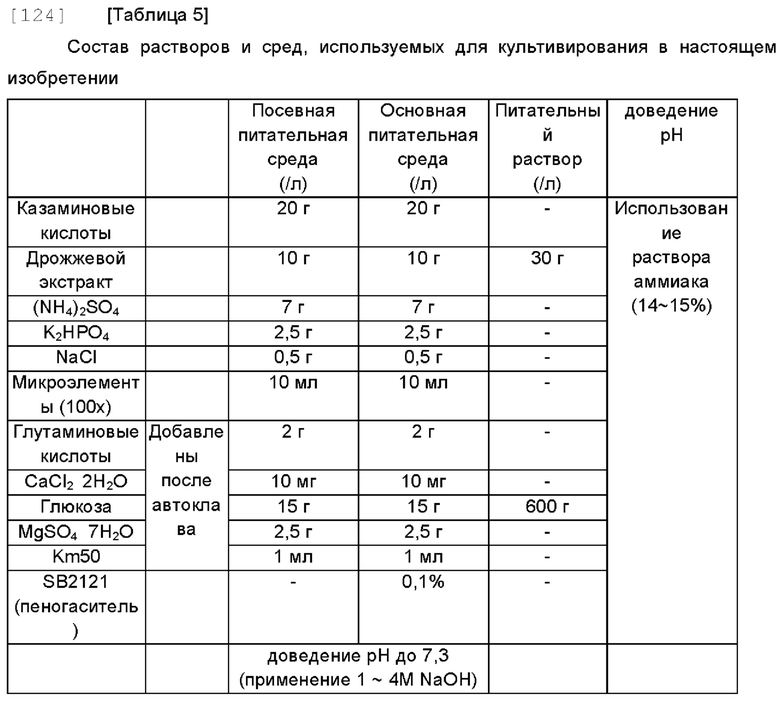
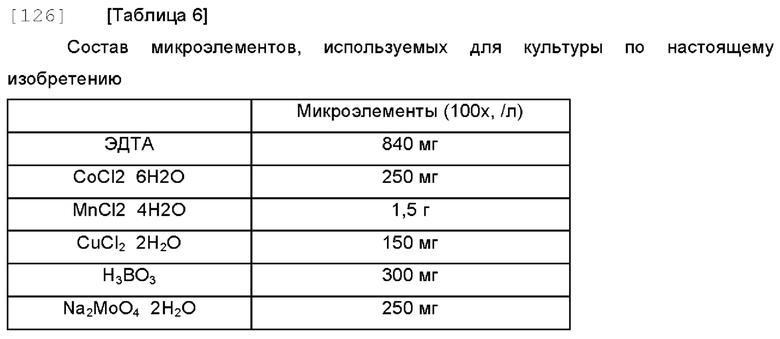
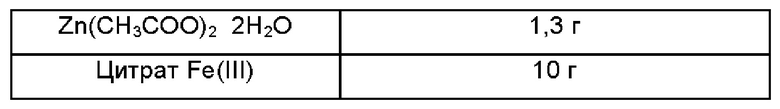
[122] Штамм BL21 (DE3), содержащий pHex-L3-CRM или pHex-L5-CRM, культивировали с использованием следующего метода. Во время основного культивирования питание осуществляли с использованием метода рН stat, а рН поддерживали на уровне 7,3 с помощью питательного раствора (600 г/л глюкозы, 30 г/л дрожжевого экстракта) и раствора щелочи (14-15% аммиака). Составы растворов и сред, используемых для культивирования, показаны в Таблицах 5 и 6.
[123]
[125]
[125]
[127]
[128] Одиночная колония, образованная в модифицированной агаровой среде LB [модифицированный агар Луриа-Бертани (LB): 10 г/л сойтона, 5 г/л дрожжевого экстракта, 10 г/л натрия хлорида, 15 г/л агара, 50 мг /л канамицина] инокулировали в посевную культуральную среду с последующей инкубацией при 30°С в течение 18 часов. Полученную посевную культуру снова инокулировали в соотношении 1% (об./об.) в основную культуральную среду (ферментер 3 л/5 л) и культивировали при 30°С. Основную питательную среду получали добавлением 0,1% стерилизованного пеногасителя к посевной питательной среде. После того как оптическая плотность культурального раствора достигала 30-40, температуру снижали до 25°С. Затем добавляли 10 мМ IPTG и культивирование прекращали после того, как поглощение культурального раствора достигало 100-120.
[12 9] Поведение культуры Е. coli BL21 (DE3), содержащей pHex-L3-CRM, в ферментере объемом 5 л показано на ФИГ. 9, а поведение культуры Е. coli BL21 (DE3), содержащей pHex-L5-CRM, показано на ФИГ. 10. Характер экспрессии CRM197 при ферментации в ферментере объемом 5 л показан на ФИГ. 11, а выход экспрессии составлял от 1,1 до 1,2 г/л для слияния с L3 и от 3,0 до 3,7 г/л для слияния с L5. Количество CRM197 измеряли путем сравнения относительного количества с эталонным CRM197 с использованием денситометра (GS-900™, Bio-Rad Laboratories Ins., Hercules, California) после окрашивания SDS-PAGE/Кумасси.
[130]
[131] Пример 4: Очистка белка
[132] Пример 4.1: Получение периплазматической фракции из клеточной культуры
[133] Процедура выделения периплазматической фракции из клеток, культивируемых в 5-литровом ферментере, выглядит следующим образом. Среду для культивирования клеток центрифугировали при 4°С и 4000 × g в течение 15 минут для осаждения клеток. Осадок клеток ресуспендировали в буфере для индукции плазматической мембраны (Таблица 7) с модифицированным белком, исходя из коэффициента поглощения 100, и перемешивали при комнатной температуре от 0,5 до 1 часа.
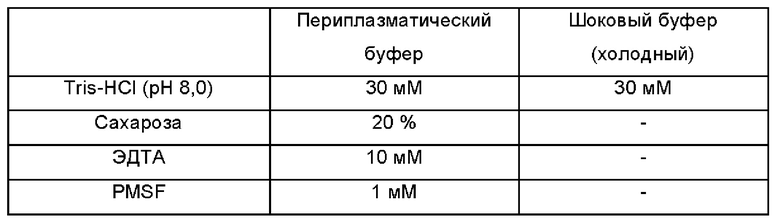
[134]

[136]
[137] Затем клетки собирали центрифугированием при 4.000 × g в течение 15 минут и добавляли такое же количество 30 мМ холодного (4°С или ниже) Tris-HCI (рН 8,0) с последующим перемешиванием при комнатной температуре в течение 0,5-1 часа. Затем клетки центрифугировали при 4000 g в течение 15 минут для получения супернатанта, и удаляли примеси с помощью MF. Анализ SDS-PAGE периплазматической фракции, извлеченной из Е, coli BL21 (DE3), содержащей рНех-L3-CRM, посредством описанного выше процесса, показан на ФИГ. 12 (А), а анализ SDS-PAGE периплазматической фракции, извлеченной из Е. coli BL21 (DE3), содержащей pHex-L5-CRM, показан на ФИГ. 12(B). Было обнаружено, что количество белка CRM197, присутствующего в периплазматической фракции, составляет 1,2 г/л для штамма L3 и 2,3 г/л для штамма L5 (Таблица 8).
[138]
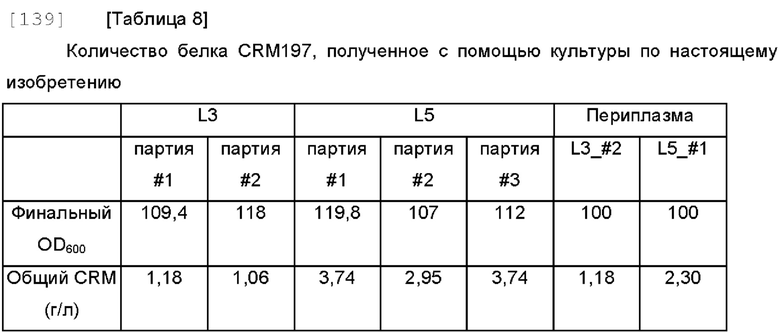
[140]
[141] Пример 4.2: Очистка белка CRM197
[142] Периплазматическую фракцию культуральной среды pHex-L3-CRM дважды концентрировали с использованием мембраны с отсечкой 10 кДа, используя систему TFF, и проводили ультрафильтрацию с использованием десяти объемов 10 мМ растворителя фосфата натрия (рН 7,2). Очистку проводили с помощью двухколоночных процессов с использованием системы AKTA pure (GE Healthcare). Первый колоночный процесс представлял собой анионообменную хроматографию (смола диэтиламиноэтилсефарозы с быстрым потоком, DEAE) и использовался для удаления нуклеиновых кислот и примесных белков. Смола DEAE заряжена отрицательно (-) и связана с положительно заряженным (+) белком. Несвязанные белки и примеси в первую очередь экстрагировали и удаляли из ультрафильтрованного образца с помощью DEAE-хроматографии, а затем неочищенные белки с низкой связывающей способностью, за исключением CRM197, удаляли с помощью последующего процесса промывки в зависимости от концентрации соли. Затем при увеличении концентрации соли элюировали только CRM197. Анализ образца в SDS-PAGE проводили во время процесса очистки с использованием DEAE-хроматографии, и результаты показаны на ФИГ. 13(A). Во-первых, несвязанные примеси и белки в первую очередь удаляли проточным способом из образца, подвергнутого DEAE-хроматографии с использованием хроматографии на гидроксиапатите (НА), и CRM197 элюировали растворителями 100 мМ фосфата калия и 100 мМ NaCI. Результат SDS-PAGE элюированного CRM197 показан на ФИГ. 13(B).
[143]
[144] Пример 5: Сравнение с CRM197, полученным с использованием Corynebacterium
[145] Были проанализированы качество и характеристики конечного очищенного CRM197. В результате анализа SEC-HPLC было обнаружено, что чистота составляла 99% или более (ФИГ. 14). SDS-PAGE и вестерн-блоттинг показали, что полоса появилась в том же положении, что и у CRM197, полученного с использованием Corynebacterium (ФИГ. 15).
[146] Кроме того, вся последовательность из 535 аминокислот, составляющих CRM197, оказалась на 100% идентичной, а в результате измерения молекулярной массы был идентифицирован основной пик 58 409 Да, что соответствовало теоретической молекулярной массе (ФИГ. 16). Структуру более высокого порядка идентифицировали с помощью анализа кругового дихроизма (CD), и было обнаружено, что не было различий в структуре более высокого порядка с CRM197, полученным с использованием Corynebacterium (ФИГ. 17). Анализ спектра флуоресценции показал, что максимальная длина волны испускания составляла 338 нм, что было таким же, как у CRM197, полученного с использованием Corynebacterium (ФИГ. 18).
[147] Результаты, описанные выше, показали, что CRM197, полученный с использованием штамма Е. coli pHex-L3, был физико-химически и иммунологически таким же, как CRM197, полученный с использованием Corynebacterium.
[148]
Промышленная применимость
[149] Настоящее изобретение очень походит для продукции белка CRM197, поскольку белок CRM197, обладающий такими же физико-химическими/иммунологическими свойствами, что и белок, выделенный из исходных бактерий, может экспрессироваться в обычной Е. coli, в которой окислительно-восстановительный потенциал не регулируется, и CRM197 белок, обладающий высокой эффективностью секреции в периплазму, может быть получен без смещения рН среды для увеличения секреции в периплазму.
[150]
[151] Несмотря на подробное описание конкретных конфигураций настоящего изобретения, специалистам в данной области техники будет понятно, что это описание представлено для изложения предпочтительных вариантов осуществления в иллюстративных целях и не должно рассматриваться как ограничивающее объем настоящего изобретения. Таким образом, существенный объем настоящего изобретения определяется прилагаемой формулой изобретения и ее эквивалентами.
[152]
Перечень последовательностей в форме свободного текста
[153] Файл в электронном виде приложен.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Genofocus, Inc.
<120> СПОСОБ ЭКСПРЕССИИ БЕЛКА CRM197
<130> P19-B209
<160> 56
<170> KoPatentIn 3.0
<210> 1
<211> 1226
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TPB1Tv1.3
<400> 1
tactgagcta ataacaggcc tgctggtaat cgcaggcctt tttatttctg gctcaccttc 60
gggtgggcct ttctgcgttt acggttctgg caaatattct gaaatgagct gttgacaatt 120
aatcatccgg ctcgtataat gtgtggaatt gtgagcggat aacaatttac tgctctttaa 180
caatttatca gatccaattg gaggaacaat atgggagacc acgcgcgcga ggtctcatct 240
gcggcggcag cagggggatc gatgagcaaa ggtgaagagc tgtttaccgg tgtggtgccg 300
attctggttg aactggatgg cgacgttaac ggccataaat tcagcgtcag cggcgagggc 360
gaaggggatg ccacctacgg taaactgacc ctgaagttta tttgcaccac cggcaaatta 420
ccggttccgt ggccgacgct ggtgacgacc tttagctatg gcgtgcagtg cttttcccgt 480
tatccggacc atatgaaaca gcatgatttt tttaaaagcg cgatgccgga aggctatgtt 540
caggaacgta ccattttctt taaggatgac ggcaattaca aaacccgcgc ggaagtgaaa 600
tttgaaggtg ataccctggt caaccgcatt gaactgaaag gcattgattt caaagaagat 660
ggtaatattc tcggtcataa gctggaatat aactacaaca gccataacgt ttatatcatg 720
gcggataaac aaaaaaacgg tattaaagtg aactttaaaa ttcgccataa tatcgaagat 780
ggtagcgtgc aactggcgga tcattatcag cagaacaccc caattggcga tggcccggtg 840
ttgctgccgg ataaccacta tctgagcacc cagtcggcgc tgagtaaaga tccgaacgaa 900
aaacgcgatc acatggtgct gctggagttt gtcaccgccg ccggtatcac ccacggcatg 960
gatgaactgt ataaataatg atttaaagcg gatatcaaat aaaacgaaag gctcagtcga 1020
aagactgggc ctttcgtttt atctgttgtt tgtcggtgaa cgctctcctg agtaggacaa 1080
atccgccggg agcggatttg aacgttgcga agcaacggcc cggagggtgg cgggcaggac 1140
gcccgccata aactgccagg catcaaatta agcagaaggc catcctgacg gatggccttt 1200
ttgcgtttct acaaactctt tttgtt 1226
<210> 2
<211> 1625
<212> ДНК
<213> Искусственная последовательность
<220>
<223> CRM197ec
<400> 2
gaagacacgg cgctgacgat gttgttgaca gcagcaagag ctttgttatg gagaatttca 60
gcagctatca cggcaccaaa ccgggctatg ttgacagcat tcagaaaggc atccaaaagc 120
cgaaaagcgg tacccagggc aactacgacg atgactggaa agagttttat agcaccgata 180
acaagtacga cgcggcgggc tatagcgttg ataacgaaaa cccgctgagc ggtaaagcgg 240
gtggcgtggt taaggtgacc tacccgggtc tgaccaaagt gctggcgctg aaggttgaca 300
acgcggaaac catcaagaaa gagctgggcc tgagcctgac cgaaccgctg atggagcagg 360
ttggtaccga ggaattcatt aagcgttttg gtgatggtgc gagccgtgtg gttctgagcc 420
tgccgttcgc ggaaggtagc agcagcgtgg agtacatcaa caactgggaa caagcgaaag 480
cgctgagcgt ggagctggaa attaacttcg aaacccgtgg caagcgtggc caggatgcga 540
tgtacgaata tatggcgcaa gcgtgcgcgg gtaaccgtgt gcgtcgtagc gttggtagca 600
gcctgagctg cattaacctg gattgggacg ttatccgtga caagaccaaa accaagatcg 660
aaagcctgaa agagcacggt ccgattaaaa acaagatgag cgagagcccg aacaagaccg 720
tgagcgagga aaaagcgaag cagtacctgg aggaatttca ccaaaccgcg ctggagcacc 780
cggaactgag cgagctgaaa accgtgaccg gcaccaaccc ggttttcgcg ggtgcgaact 840
atgcggcgtg ggcggtgaac gttgcgcagg tgatcgatag cgaaaccgcg gacaacctgg 900
aaaagaccac cgcggcgctg agcatcctgc cgggtattgg cagcgtgatg ggcatcgcgg 960
atggtgcggt tcaccacaac accgaggaaa tcgtggcgca gagcattgcg ctgagcagcc 1020
tgatggttgc gcaagcgatc ccgctggttg gtgagctggt tgacattggt ttcgcggcgt 1080
acaactttgt ggaaagcatc attaacctgt tccaggtggt tcacaacagc tacaaccgtc 1140
cggcgtatag cccgggccac aaaacccaac cgtttctgca cgatggttat gcggtgagct 1200
ggaacaccgt tgaggacagc atcattcgta ccggtttcca gggcgagagc ggtcacgata 1260
tcaagattac cgcggaaaac accccgctgc cgattgcggg cgttctgctg ccgaccattc 1320
cgggtaaact ggacgttaac aaaagcaaga cccacattag cgtgaacggc cgtaagatcc 1380
gtatgcgttg ccgtgcgatt gatggtgacg tgaccttttg ccgtccgaaa agcccggtgt 1440
acgttggtaa cggcgtgcac gcgaacctgc acgttgcgtt ccaccgtagc agcagcgaga 1500
agatccacag caacgaaatt agcagcgaca gcatcggcgt tctgggttat caaaaaaccg 1560
tggatcatac caaagtgaat agcaagctga gcctgttctt cgagattaaa agctctgagg 1620
tcttc 1625
<210> 3
<211> 535
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CRM197
<400> 3
Gly Ala Asp Asp Val Val Asp Ser Ser Lys Ser Phe Val Met Glu Asn
1 5 10 15
Phe Ser Ser Tyr His Gly Thr Lys Pro Gly Tyr Val Asp Ser Ile Gln
20 25 30
Lys Gly Ile Gln Lys Pro Lys Ser Gly Thr Gln Gly Asn Tyr Asp Asp
35 40 45
Asp Trp Lys Glu Phe Tyr Ser Thr Asp Asn Lys Tyr Asp Ala Ala Gly
50 55 60
Tyr Ser Val Asp Asn Glu Asn Pro Leu Ser Gly Lys Ala Gly Gly Val
65 70 75 80
Val Lys Val Thr Tyr Pro Gly Leu Thr Lys Val Leu Ala Leu Lys Val
85 90 95
Asp Asn Ala Glu Thr Ile Lys Lys Glu Leu Gly Leu Ser Leu Thr Glu
100 105 110
Pro Leu Met Glu Gln Val Gly Thr Glu Glu Phe Ile Lys Arg Phe Gly
115 120 125
Asp Gly Ala Ser Arg Val Val Leu Ser Leu Pro Phe Ala Glu Gly Ser
130 135 140
Ser Ser Val Glu Tyr Ile Asn Asn Trp Glu Gln Ala Lys Ala Leu Ser
145 150 155 160
Val Glu Leu Glu Ile Asn Phe Glu Thr Arg Gly Lys Arg Gly Gln Asp
165 170 175
Ala Met Tyr Glu Tyr Met Ala Gln Ala Cys Ala Gly Asn Arg Val Arg
180 185 190
Arg Ser Val Gly Ser Ser Leu Ser Cys Ile Asn Leu Asp Trp Asp Val
195 200 205
Ile Arg Asp Lys Thr Lys Thr Lys Ile Glu Ser Leu Lys Glu His Gly
210 215 220
Pro Ile Lys Asn Lys Met Ser Glu Ser Pro Asn Lys Thr Val Ser Glu
225 230 235 240
Glu Lys Ala Lys Gln Tyr Leu Glu Glu Phe His Gln Thr Ala Leu Glu
245 250 255
His Pro Glu Leu Ser Glu Leu Lys Thr Val Thr Gly Thr Asn Pro Val
260 265 270
Phe Ala Gly Ala Asn Tyr Ala Ala Trp Ala Val Asn Val Ala Gln Val
275 280 285
Ile Asp Ser Glu Thr Ala Asp Asn Leu Glu Lys Thr Thr Ala Ala Leu
290 295 300
Ser Ile Leu Pro Gly Ile Gly Ser Val Met Gly Ile Ala Asp Gly Ala
305 310 315 320
Val His His Asn Thr Glu Glu Ile Val Ala Gln Ser Ile Ala Leu Ser
325 330 335
Ser Leu Met Val Ala Gln Ala Ile Pro Leu Val Gly Glu Leu Val Asp
340 345 350
Ile Gly Phe Ala Ala Tyr Asn Phe Val Glu Ser Ile Ile Asn Leu Phe
355 360 365
Gln Val Val His Asn Ser Tyr Asn Arg Pro Ala Tyr Ser Pro Gly His
370 375 380
Lys Thr Gln Pro Phe Leu His Asp Gly Tyr Ala Val Ser Trp Asn Thr
385 390 395 400
Val Glu Asp Ser Ile Ile Arg Thr Gly Phe Gln Gly Glu Ser Gly His
405 410 415
Asp Ile Lys Ile Thr Ala Glu Asn Thr Pro Leu Pro Ile Ala Gly Val
420 425 430
Leu Leu Pro Thr Ile Pro Gly Lys Leu Asp Val Asn Lys Ser Lys Thr
435 440 445
His Ile Ser Val Asn Gly Arg Lys Ile Arg Met Arg Cys Arg Ala Ile
450 455 460
Asp Gly Asp Val Thr Phe Cys Arg Pro Lys Ser Pro Val Tyr Val Gly
465 470 475 480
Asn Gly Val His Ala Asn Leu His Val Ala Phe His Arg Ser Ser Ser
485 490 495
Glu Lys Ile His Ser Asn Glu Ile Ser Ser Asp Ser Ile Gly Val Leu
500 505 510
Gly Tyr Gln Lys Thr Val Asp His Thr Lys Val Asn Ser Lys Leu Ser
515 520 525
Leu Phe Phe Glu Ile Lys Ser
530 535
<210> 4
<211> 85
<212> ДНК
<213> Искусственная последовательность
<220>
<223> PelB (L1)
<400> 4
ggtctcatat gaaatatctg ttaccgaccg ccgctgccgg actgctgtta ctggcggcgc 60
agccggcgat ggcgggcgag agacc 85
<210> 5
<211> 88
<212> ДНК
<213> Искусственная последовательность
<220>
<223> G8 (L2)
<400> 5
ggtctcatat gaaaaaaagc ctggttctga aagcgtctgt tgcggtggcg acgctggtgc 60
cgatgctgtc gtttgccggc gagagacc 88
<210> 6
<211> 73
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Wp (L3)
<400> 6
ggtctcatat gcgttctgtg attgttgcct tcctgtttgc ctgtagcttt tgcgtgagcg 60
ccggcgagag acc 73
<210> 7
<211> 79
<212> ДНК
<213> Искусственная последовательность
<220>
<223> OmpT (L4)
<400> 7
ggtctcatat gcgtgcgaaa ctgctcggca ttgttctgac caccccgatt gccatttcca 60
gctttgccgg cgagagacc 79
<210> 8
<211> 82
<212> ДНК
<213> Искусственная последовательность
<220>
<223> OmpA (L5)
<400> 8
ggtctcatat gaaaaaaacc gccatcgcca ttgccgttgc cctcgctggc tttgccaccg 60
tggcgcaggc gggcgagaga cc 82
<210> 9
<211> 82
<212> ДНК
<213> Искусственная последовательность
<220>
<223> G4 (L6)
<400> 9
ggtctcatat gaaactgctg aacgtgatca actttgtttt cctgatgttt gtcagcagca 60
gtagttttgc cggcgagaga cc 82
<210> 10
<211> 73
<212> ДНК
<213> Искусственная последовательность
<220>
<223> G3 (L7)
<400> 10
ggtctcatat gaaaaaactg ctgtttgcca ttccgctggt tgtaccgttt tacagccaca 60
gcggcgagag acc 73
<210> 11
<211> 79
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Lpp (L8)
<400> 11
ggtctcatat gaaagcgacg aaactggtgc tgggtgctgt gattctgggc agcacgctgc 60
tggcgggcgg cgagagacc 79
<210> 12
<211> 88
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Gsp (L9)
<400> 12
ggtctcatat gaaaggtctg aataaaatta cctgctgttt actggcggcg ctgctgatgc 60
cgtgcgcggg tcatgcgggc gagagacc 88
<210> 13
<211> 22
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> PelB (L1)
<400> 13
Met Lys Tyr Leu Leu Pro Thr Ala Ala Ala Gly Leu Leu Leu Leu Ala
1 5 10 15
Ala Gln Pro Ala Met Ala
20
<210> 14
<211> 23
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> G8 (L2)
<400> 14
Met Lys Lys Ser Leu Val Leu Lys Ala Ser Val Ala Val Ala Thr Leu
1 5 10 15
Val Pro Met Leu Ser Phe Ala
20
<210> 15
<211> 18
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Wp (L3)
<400> 15
Met Arg Ser Val Ile Val Ala Phe Leu Phe Ala Cys Ser Phe Cys Val
1 5 10 15
Ser Ala
<210> 16
<211> 20
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> OmpT (L4)
<400> 16
Met Arg Ala Lys Leu Leu Gly Ile Val Leu Thr Thr Pro Ile Ala Ile
1 5 10 15
Ser Ser Phe Ala
20
<210> 17
<211> 21
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> OmpA (L5)
<400> 17
Met Lys Lys Thr Ala Ile Ala Ile Ala Val Ala Leu Ala Gly Phe Ala
1 5 10 15
Thr Val Ala Gln Ala
20
<210> 18
<211> 21
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> G4 (L6)
<400> 18
Met Lys Leu Leu Asn Val Ile Asn Phe Val Phe Leu Met Phe Val Ser
1 5 10 15
Ser Ser Ser Phe Ala
20
<210> 19
<211> 18
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> G3 (L7)
<400> 19
Met Lys Lys Leu Leu Phe Ala Ile Pro Leu Val Val Pro Phe Tyr Ser
1 5 10 15
His Ser
<210> 20
<211> 20
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Lpp (L8)
<400> 20
Met Lys Ala Thr Lys Leu Val Leu Gly Ala Val Ile Leu Gly Ser Thr
1 5 10 15
Leu Leu Ala Gly
20
<210> 21
<211> 23
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Gsp (L9)
<400> 21
Met Lys Gly Leu Asn Lys Ile Thr Cys Cys Leu Leu Ala Ala Leu Leu
1 5 10 15
Met Pro Cys Ala Gly His Ala
20
<210> 22
<211> 34
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 22
gcggatccaa gagacaggat gaggatcgtt tcgc 34
<210> 23
<211> 40
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 23
cggatatcaa gcttggaaat gttgaatact catactcttc 40
<210> 24
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 24
gagatccgga gcttatactg agctaataac 30
<210> 25
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 25
gaaaaataaa caaaaacaaa aagagtttg 29
<210> 26
<211> 31
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 26
tacaaactct ttttgttttt gtttattttt c 31
<210> 27
<211> 34
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 27
cctgttatta gctcagtata agctccggat ctcg 34
<210> 28
<211> 26
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 28
aattggagga acaatatgaa atatct 26
<210> 29
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 29
aacatcgtca gcgcccgcca tcgccggct 29
<210> 30
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 30
ttggaggaac aatatgaaaa aaagcct 27
<210> 31
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 31
aacatcgtca gcgccggcaa acgacagcat 30
<210> 32
<211> 26
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 32
ttggaggaac aatatgcgtt ctgtga 26
<210> 33
<211> 28
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 33
aacatcgtca gcgccggcgc tcacgcaa 28
<210> 34
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 34
ttggaggaac aatatgcgtg cgaaact 27
<210> 35
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 35
aacatcgtca gcgccggcaa agctggaaat 30
<210> 36
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 36
ttggaggaac aatatgaaaa aaacc 25
<210> 37
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 37
aacatcgtca gcgcccgcct gcgccacggt 30
<210> 38
<211> 26
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 38
ttggaggaac aatatgaaac tgctga 26
<210> 39
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 39
aacatcgtca gcgccggcaa aactactgct 30
<210> 40
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 40
ttggaggaac aatatgaaaa aactg 25
<210> 41
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 41
acatcgtcag cgccgctgtg gctgtaaaa 29
<210> 42
<211> 28
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 42
ttggaggaac aatatgaaag cgacgaaa 28
<210> 43
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 43
aacatcgtca gcgccgcccg ccagcagcgt 30
<210> 44
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 44
ttggaggaac aatatgaaag gtctgaa 27
<210> 45
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 45
aacatcgtca gcgcccgcat gacccgcgca 30
<210> 46
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 46
agccggcgat ggcgggcgct gacgatg 27
<210> 47
<211> 28
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 47
atgctgtcgt ttgccggcgc tgacgatg 28
<210> 48
<211> 26
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 48
ttgcgtgagc gccggcgctg acgatg 26
<210> 49
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 49
tttccagctt tgccggcgct gacgatg 27
<210> 50
<211> 28
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 50
accgtggcgc aggcgggcgc tgacgatg 28
<210> 51
<211> 28
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 51
agcagtagtt ttgccggcgc tgacgatg 28
<210> 52
<211> 28
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 52
ttttacagcc acagcggcgc tgacgatg 28
<210> 53
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 53
tgctggcggg cggcgctgac gatg 24
<210> 54
<211> 26
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 54
cgcgggtcat gcgggcgctg acgatg 26
<210> 55
<211> 36
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 55
gatatccgct tttcattagc ttttaatctc gaagaa 36
<210> 56
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 56
ggcgcaagcg tgcgcgggta accgtgtgcg 30
<---


Изобретение относится к биотехнологии. Предложена конструкция нуклеиновой кислоты для экспрессии белка CRM197, содержащая нуклеиновую кислоту, кодирующую сигнальную последовательность, которая содержит последовательность SEQ ID NO: 6 или SEQ ID NO: 8, и ген белка CRM197. Также предложены вектор экспрессии для экспрессии белка CRM197, содержащий указанную конструкцию нуклеиновой кислоты, рекомбинантный микроорганизм для экспрессии белка CRM197, содержащий указанную конструкцию нуклеиновой кислоты или указанный вектор экспрессии, а также способ получения белка CRM197. Изобретение обеспечивает эффективную экспрессию белка CRM197 и эффективное секретирование белка CRM197 в периплазму без изменения рН среды. 4 н. и 5 з.п. ф-лы, 18 ил., 8 табл., 5 пр.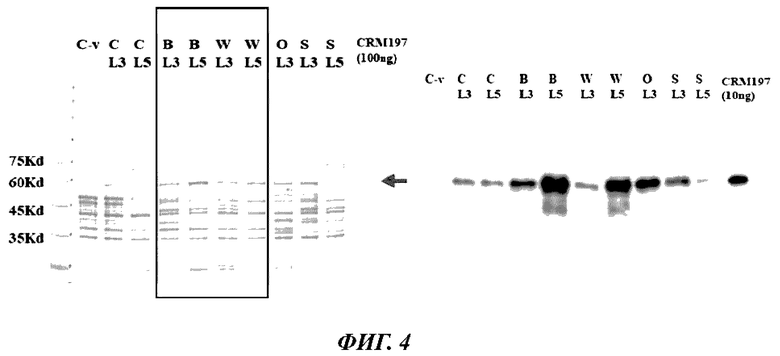
1. Конструкция нуклеиновой кислоты для экспрессии белка CRM197, содержащая нуклеиновую кислоту, кодирующую сигнальную последовательность, которая содержит последовательность SEQ ID NO: 6 или SEQ ID NO: 8, и ген белка CRM197.
2. Конструкция нуклеиновой кислоты по п.1, где указанная нуклеиновая кислота представлена нуклеотидной последовательностью SEQ ID NO: 8.
3. Конструкция нуклеиновой кислоты по п. 1, отличающаяся тем, что ген белка CRM197 представлен нуклеотидной последовательностью SEQ ID NO: 2.
4. Вектор экспрессии для экспрессии белка CRM197, содержащий конструкцию нуклеиновой кислоты по п. 1.
5. Вектор экспрессии по п. 4, дополнительно содержащий промотор Trc.
6. Рекомбинантный микроорганизм для экспрессии белка CRM197, содержащий конструкцию нуклеиновой кислоты по п. 1 или вектор экспрессии по п. 4.
7. Рекомбинантный микроорганизм по п. 6, отличающийся тем, что указанный рекомбинантный микроорганизм представляет собой Escherichia coli.
8. Способ получения белка CRM197, включающий:
(а) культивирование рекомбинантного микроорганизма по п. 6 с получением белка CRM197; и
(b) выделение полученного белка CRM197.
9. Способ по п. 8, отличающийся тем, что стадия (b) включает выделение белка CRM197, секретированного в периплазму.
| WO 2015134402 A1, 11.09.2015 | |||
| EP 3087088 A2, 02.11.2016 | |||
| Номографический прибор | 1935 |
|
SU45758A1 |
| Putative secretin GspD | |||
| Номографический прибор | 1935 |
|
SU45758A1 |
| Переносный механический струг для дерева | 1923 |
|
SU3664A1 |
| Virion export protein | |||
| Найдено онлайн: | |||
Авторы
Даты
2023-09-22—Публикация
2019-10-02—Подача