Область техники
Настоящее изобретение относится к слитому белку, посредством которого можно модифицировать митохондрии, митохондриям, модифицированным посредством слитого белка, и фармацевтической композиции, содержащей их в качестве активного ингредиента.
Предшествующий уровень техники
Митохондрии являются клеточными органеллами эукариотических клеток, участвующими в синтезе и регуляции аденозинтрифосфата (АТФ), внутриклеточного источника энергии. Митохондрии ассоциированы с различными метаболическими путями in vivo, например, передачей сигнала в клетке, дифференцировкой клеток, гибелью клеток, а также контролем клеточного цикла и роста клеток. Митохондрии имеют свои собственные геномы и являются органеллами, играющими ключевую роль в энергетическом метаболизме клеток. Митохондрии продуцируют энергию через транспорт электронов и процесс окислительного фосфорилирования и играют важную роль в апоптотических путях передачи сигнала.
Описано, что снижение продукции энергии из-за снижения функции митохондрий вызывает различные заболевания. Когда функционирование цепи транспорта электронов снижается в соответствии с изменениями генома и протеома митохондрий, происходит снижение продукции АТФ, избыточная продукция активных форм кислорода, снижение функции регуляции кальция и т.п. В этом случае происходит изменение проницаемости мембраны митохондрий, функция апоптоза может выполняться с отклонениями, что приводит к развитию злокачественных новообразований и неизлечимых заболеваний.
В связи с этим, заболевания человека, которые, как описано, являются результатом дисфункции митохондрий, включают связанные с митохондриями генетические заболевания (Wallace DC, 1999), сахарный диабет (Maechler P, 2001), заболевания сердца (Sorescu D, 2002), старческую деменцию, такую как болезнь Паркинсона или болезнь Альцгеймера(Lin MT, 2006), различные злокачественные новообразования (Petros JA, 2005), метастазирование злокачественных новообразований (Ishikawa K, 2008) и т.п. Кроме того, признаки, часто обнаруживаемые в более чем 200 типах различных злокачественных новообразований, состоят из нарушения функции апоптоза, повышенного воспалительного ответа повышенного аномального метаболизма. Все эти процессы тесно связаны с функцией митохондрий, и корреляция между злокачественными новообразованиями и митохондриями привлекает все больше внимания.
С другой стороны, известно, что нормальные клетки продуцируют 36 АТФ на молекулу глюкозы с помощью системы транспорта электронов, но злокачественные клетки, в отличие от нормальных клеток, продуцируют 2 АТФ на молекулу глюкозы посредством гликолиза в условиях достаточного количества кислорода (аэробный гликолиз). В связи с этим, известно, что злокачественные клетки, в отличие от нормальных клеток, используют неэффективный процесс гликолиза в терминах энергии для продуцирования аминокислот, липидов, нуклеиновых кислот и т.п., необходимых для быстрой пролиферация клеток. В связи с этим, известно, что злокачественным клеткам необходимо меньше кислорода, и они продуцируют больше молочной кислоты, чем нормальные клетки.
Таким образом, изменение композиции микроокружения опухоли из-за аномального метаболизма, возникающее в злокачественных клетках, ингибирование апоптоза, вызванное дисфункциональными митохондриями, повышение воспалительного ответа и аномальная метаболическая реакция в злокачественных клетках играют очень важную роль в пролиферации злокачественного новообразования. Таким образом, разработка связанных с метаболизмом противоопухолевых средств с использованием этих признаков может являться хорошим путем разрешения проблем побочных эффектов и экономических проблем общепринятых противоопухолевых средств.
Известно, что митохондрии проникают в клетки, когда митохондрии, присутствующие в клетках, выделяют и клетки обрабатывают ими in vitro, или митохондрии инъецируют в организм. Используя это явление, можно инъецировать нормальные митохондрии, выделенные из клеток, в организм для лечения заболеваний, вызванных дисфункцией митохондрий или, в частности, для лечения заболеваний посредством эффективной доставки конкретного белка в клетки с использованием митохондрий в качестве носителя, но это еще не описано.
Техническая задача
Целью настоящего изобретения является предоставление эффективной системы доставки белка посредством демонстрации того, что митохондрии можно использовать в качестве средств для эффективной доставки белков, способных проявлять различные фармакологические эффекты в клетках. Кроме того, целью настоящего изобретения является предоставление рекомбинантного белка для эффективной доставки лекарственного средства и для получения модифицированных митохондрий, получаемых с помощью него. Кроме того, целью настоящего изобретения является предоставление фармацевтической композиции, содержащей модифицированные митохондрии в качестве активного ингредиента.
Решение технической задачи
Что касается решения указанных выше задач, настоящее изобретение относится к модифицированным митохондриям, в которых чужеродный белок связан с внешней мембраной митохондрий. Кроме того, что касается получения модифицированных митохондрий, настоящее изобретение относится к слитому белку, содержащему пептид, заякоривающий в митохондриальной внешней мембране, и желаемый фармакологический белок. Кроме того, настоящее изобретение относится к слитому белку, содержащему антитело или его фрагмент и пептид, заякоривающий в митохондриальной внешней мембране.
Эффект изобретения
Чужеродный белок можно эффективно доставлять в клетку, если митохондрии, с которыми связан чужеродный белок, вводят в организм человека. Кроме того, с помощью фармакологически активного белка, доставляемого в клетку, можно восстанавливать нарушенную функцию клеток. Кроме того, если митохондрии, с которыми связан чужеродный белок, содержащий фармакологически активный белок, доставляют в клетку, фармакологически активный белок диссоциирует от митохондрий в клетке, и можно ожидать, что он будет выполнять полезную роль. Кроме того, в клетки-мишени можно эффективно доставлять модифицированные митохондрии, содержащие фрагмент антитела. В частности, если фрагмент антитела против белка, присутствующего на поверхности злокачественной ткани, связан с поверхностью митохондрий, модифицированные митохондрии можно эффективно доставлять в злокачественные клетки. Таким образом, посредством введения модифицированных митохондрий не только можно восстанавливать поврежденную систему транспорта электронов клеток, но также можно и предотвращать или лечить различные заболевания с помощью фармакологически активного белка, связанного с модифицированными митохондриями.
Краткое описание чертежей
Фигура 1 является схемой способа получения pTA-p53.
Фигура 2 является схемой способа получения вектора pET15b-UB-p53.
На фигуре 3 показана экспрессия белка UB-p53 в E. coli.
Фигура 4 является схемой способа получения вектора pET11C-TOM70-UB-p53.
На фигуре 5 показана экспрессия белка TOM70-UB-p53 в E. coli.
Фигура 6 является схемой способа получения вектора pET11C-TOM70-(GGGGS)3-UB-p53.
На фигуре 7 показана экспрессия белка TOM70-(GGGGS)3-UB-p53 в E. coli.
На фигуре 8 показан способ получения вектора pET11C-TOM70-(GGGGS)3-p53.
На фигуре 9 показана экспрессия белка TOM70-(GGGGS)3-p53 в E. coli.
На фигуре 10 показан способ получения вектора pET15b-UB-p53-TOM7.
На фигуре 11 показана экспрессия белка UB-p53-TOM7 в E. coli.
На фигуре 12 показан способ получения вектора pCMV-p53-myc/His.
На фигуре 13 показана экспрессия белка p53-myc/His в трансформированных CHO.
На фигуре 14 показаны результаты очистки белка TOM70-(GGGGS)3-p53, а затем его идентификации.
На фигуре 15 показан очищенный белок TOM70-(GGGGS)3-p53.
На фигуре 16 показаны результаты очистки белка TOM70-(GGGGS)3-UB-p53, а затем его идентификации.
На фигуре 17 показан очищенный белок TOM70-(GGGGS)3-UB-p53.
На фигуре 18 показаны результаты очистки белка UB-p53, а затем его идентификации.
На фигуре 19 показан очищенный белок UB-p53.
На фигуре 20 показаны результаты очистки белка UB-p53-TOM7, а затем его идентификации.
На фигуре 21 показан очищенный белок UB-p53-TOM7.
На фигуре 22 показан способ получения вектора pTA-гранзим B.
На фигуре 23 показан способ получения вектора pET11C-TOM70-(GGGGS)3-UB-гранзим B.
На фигуре 24 показана экспрессия белка TOM70-(GGGGS)3-UB-гранзим B в E. coli.
На фигуре 25 показан способ получения вектора pET15b-UB-гранзим B-TOM7.
На фигуре 26 показана экспрессия белка UB-гранзим B-TOM7 в E. coli.
На фигуре 27 показаны результаты очистки белка TOM70-(GGGGS)3-UB-гранзим B.
На фигуре 28 показан очищенный белок TOM70-(GGGGS)3-UB-гранзим B.
На фигуре 29 показан способ получения вектора pTA-RKIP.
На фигуре 30 показан способ получения вектора pET11C-TOM70-(GGGGS)3-UB-RKIP.
На фигуре 31 показана экспрессия белка TOM70-(GGGGS)3-UB-RKIP в E. coli.
На фигуре 32 показаны результаты очистки белка TOM70-(GGGGS)3-UB-RKIP.
На фигуре 33 показан очищенный белок TOM70-(GGGGS)3-UB-RKIP.
На фигуре 34 показан способ получения вектора pTA-PTEN.
На фигуре 35 показан способ получения вектора pET11C-TOM70-(GGGGS)3-UB-PTEN.
На фигуре 36 показана экспрессия белка TOM70-(GGGGS)3-UB-PTE в E. coli.
На фигуре 37 показаны результаты очистки белка TOM70-(GGGGS)3-UB-PTEN.
На фигуре 38 показан очищенный белок TOM70-(GGGGS)3-UB-PTEN.
На фигуре 39 показаны результаты очистки белка UB-GFP-TOM7, а затем его идентификации.
На фигуре 40 показан очищенный белок UB-GFP-TOM7.
На фигуре 41 показаны результаты очистки белка TOM70-(GGGGS)3-UB-GFP, а затем его идентификации.
На фигуре 42 показан очищенный белок TOM70-(GGGGS)3-UB-GFP.
На фигуре 43 показан способ получения вектора pET15b-UB-scFvHER2-TOM7.
На фигуре 44 показана экспрессия белка UB-scFvHER2-TOM7 в E. coli.
На фигуре 45 показан способ получения вектора pCMV-scFvHER2-TOM7-myc/His.
На фигуре 46 показана экспрессия белка scFvHER2-TOM7-myc/His в трансформированных CHO.
На фигуре 47 показаны результаты очистки белка UB-ScFvHER2-TOM7.
На фигуре 48 показан очищенный белок UB-ScFvHER2-TOM7.
На фигуре 49 показан способ получения вектора pET15b-UB-scFvMEL-TOM7.
На фигуре 50 показана экспрессия белка UB-scFvMEL-TOM7 в E. coli.
На фигуре 51 показан способ получения вектора pCMV-scFvMEL-TOM7-myc/His.
На фигуре 52 показана экспрессия белка scFvMEL-TOM7-myc/His в трансформированных CHO.
На фигуре 53 показан способ получения вектора pCMV-scFvPD-L1-TOM7-myc/His.
На фигуре 54 показана экспрессия белка scFvPD-L1-TOM7-myc/His в трансформированных CHO.
Фигура 55 является изображением, на котором подтверждено, что флуоресцентный белок связан с внешней мембраной митохондрий. В этом случае митохондрии окрашивают с помощью MitoTracker CMXRos для демонстрации красного окрашивания и TOM70-UB-GFP для демонстрации зеленого окрашивания. Область перекрывания двух частей демонстрирует желтое окрашивание. В этом случае увеличение на 55a является 200-кратным, и увеличение на 55b является 600-кратным.
На фигуре 56 показаны результаты идентификации рекомбинантного белка TOM70-(GGGGS)3-UB-p53 и UB-p53-TOM7, связанного с внешней мембраной чужеродных митохондрий, посредством анализа вестерн-блоттинга.
На фигуре 57 показаны результаты наблюдения степени внутриклеточной инъекции в соответствии с концентрацией митохондрий с использованием флуоресцентного микроскопа после выделения чужеродных митохондрий, а затем инъекции митохондрий в клетки.
Фигура 58 является изображением, на котором подтверждено влияние нормальных митохондрий на пролиферацию злокачественных клеток кожи.
Фигура 59 является изображением, на котором подтверждено влияние нормальных митохондрий на ингибирование продукции активных форм кислорода (ROS) в злокачественных клетках кожи.
Фигура 60 является изображением, на котором подтверждено влияние нормальных митохондрий на резистентность к лекарственным средствам.
Фигура 61 является изображением, на котором подтверждено влияние нормальных митохондрий на экспрессию генов антиоксидантов в клетках.
Фигура 62 является изображением, на котором показано влияние нормальных митохондрий на экспрессию генов, участвующих в метастазировании злокачественных клеток.
Фигура 63 является схемой способа подтверждения нагрузки рекомбинантного белка p53 на внешнюю мембрану чужеродных митохондрий и инъекции рекомбинантного белка p53 в клетку.
Фигура 64 является изображением, на котором подтверждено, что рекомбинантный белок p53 нагружают на внешнюю мембрану чужеродных митохондрий, и что p53 инъецируют в клетку. В этом случае увеличение является 200-кратным.
Фигура 65 является изображением, на котором подтверждено, что рекомбинантный белок p53 нагружают на внешнюю мембрану чужеродных митохондрий, и что p53 инъецируют в клетку. В этом случае увеличение является 600-кратным.
Фигура 66 является схемой способа подтверждения апоптотической способности модифицированных митохондрий, на которые нагружают p53, инъецируемый в клетки, с использованием линии клеток рака желудка.
Фигура 67a является изображением, на котором подтверждена апоптотическая способность модифицированных митохондрий, на которые нагружен p53, инъецированный в клетки рака желудка, посредством анализа TUNEL. В этом случае увеличение является 600-кратным.
Фигура 67b является изображением, на котором подтверждена апоптотическая способность модифицированных митохондрий, на которые нагружен p53, инъецированный в клетки рака желудка, посредством измерения флуоресценции.
Фигура 68 является изображением, на котором подтвержден эффект ингибирования метастазирования злокачественных клеток с помощью модифицированных митохондрий, нагруженных RKIP, в клетках MDA-MB-231.
Фигура 69 является изображением, на котором подтверждено, что в клетках экспрессируется одноцепочечный вариабельный фрагмент (ScFv) антитела для таргетинга злокачественных клеток.
Фигура 70 является изображением, на котором с использованием экспериментального способа иммуноцитохимии (ICC) подтверждено, что одноцепочечный вариабельный фрагмент (ScFv) антитела для таргетинга злокачественных клеток экспрессируется и связан с митохондриями, присутствующими в клетке. В этом случае увеличение является 200-кратным.
Фигура 71 является изображением, на котором с использованием экспериментального способа иммуноцитохимии (ICC) подтверждено, что одноцепочечный вариабельный фрагмент (ScFv) антитела для таргетинга злокачественных клеток экспрессируется и связан с митохондриями, присутствующими в клетке. В этом случае увеличение является 600-кратным.
Фигура 72 является изображением, на котором сравнивают эффект инъекции митохондрий, с которыми связан одноцепочечный вариабельный фрагмент антитела для таргетинга злокачественных клеток, в линии клеток рака желудка.
Фигура 73 является схемой эксперимента на животных с использованием модифицированных митохондрий.
Фигура 74 является фотографией, на которой видно увеличение опухолевой ткани.
Фигура 75 является изображением, на котором подтверждено изменение массы тела мышей после введения митохондрий и модифицированных митохондрий.
Фигура 76 является изображением, на котором подтвержден размер опухоли после введения митохондрий и модифицированных митохондрий.
Фигура 77 является изображением, на котором подтверждено, что модифицированные митохондрии, нагруженные белком TOM-UB-p53, являются эффективными в ингибировании пролиферации клеток A431.
Фигура 78 является изображением, на котором подтверждена функция выделенных митохондрий в отношении содержания АТФ.
Фигура 79 является изображением, на котором подтверждена функция выделенных митохондрий в отношении мембранного потенциала.
Фигура 80 является изображением, на котором подтверждена степень повреждения выделенных митохондрий посредством измерения митохондриальных ROS (продукции mROS).
Фигура 81a является изображением, на котором показана структура белка, присутствующего во внешней мембране митохондрий, и аминокислотная последовательность N-концевой области TOM70, TOM20 или OM45.
Фигура 81b является изображением, на котором показана аминокислотная последовательность C-концевой области TOM5, TOM7, Fis1, VAMP1B, Cytb5, BCL-2 или BCL-X.
Фигура 82 является изображением, на котором подтверждено, что желаемый белок диссоциирует в зависимости от присутствия или отсутствия линкера между пептидом, заякоривающим во внешней мембране, и убиквитином.
Фигура 83 является изображением, на котором подтверждено, что желаемый белок, связанный с модифицированными митохондриями, отделен от митохондрий в клетке.
Лучший способ осуществления изобретения
Далее в настоящем описании настоящее изобретение будет описано более подробно.
В одном из аспектов настоящее изобретение относится к модифицированным митохондриям, в которых чужеродный белок связан с внешней мембраной митохондрий.
Митохондрии можно получать из млекопитающих, в частности, их можно получать из людей. В частности, митохондрии можно выделять из клеток или тканей. Например, митохондрии можно получать из соматических клеток, половых клеток или стволовых клеток. Кроме того, митохондрии могут являться нормальными митохондриями, полученными из клеток, в которых биологическая активность митохондрий является нормальной. Кроме того, митохондрии можно культивировать in vitro.
Кроме того, митохондрии можно получать из аутологичного, аллогенного или ксеногенного индивидуума. В частности, термин "аутологичные митохондрии" относится к митохондриям, полученным из тканей или клеток того же индивидуума. Кроме того, термин "аллогенные митохондрии" относится к митохондриям, полученным из индивидуума, принадлежащего к тому же биологическому виду, что и индивидуум, и имеет другие генотипы аллелей. Кроме того, термин "ксеногенные митохондрии" относится к митохондриям, полученным из индивидуума, принадлежащего к иному биологическому виду, чем индивидуум.
В частности, соматические клетки могут являться мышечными клетками, гепатоцитами, нервными клетками, фибробластами, эпителиальными клетками, адипоцитами, остеоцитами, лейкоцитами, лимфоцитами, тромбоцитами или клетками слизистых оболочек. Кроме того, половые клетки являются клетками, подвергающимися мейозу и митозу, и могут представлять собой сперматозоиды или яйцеклетки. Кроме того, стволовые клетки могут быть выбраны из группы, состоящей из мезенхимальных стволовых клеток, стволовых клеток взрослых, индуцированных плюрипотентных стволовых клеток, эмбриональных стволовых клеток, стволовых клеток костного мозга, нервных стволовых клеток, лимбальных стволовых клеток, и тканевых стволовых клеток. В этом случае мезенхимальные стволовые клетки могут быть выбраны из группы, состоящей из пуповины, пуповинной крови, костного мозга, жира, мышц, нервов, кожи, амниотической мембраны и плаценты.
С другой стороны, если митохондрии выделены из конкретных клеток, митохондрии можно выделять различными известными способами, например, с использованием конкретного буферного раствора или с использованием разницы потенциалов и магнитного поля и т.п.
В рамках изобретения термин "чужеродный белок" относится к белку, включающему желаемый белок, способный функционировать внутри и вне клетки. В этом случае чужеродный белок является белком, не существующим в митохондриях, и может являться рекомбинантным белком. В частности, чужеродный белок может содержать пептид, заякоривающий в митохондриях, и желаемый белок. Кроме того, чужеродный белок может являться рекомбинантным слитым белком, содержащим пептид, заякоривающий в митохондриях, и желаемый белок. В этом случае чужеродный белок может содержать пептид, заякоривающий в митохондриях. Предпочтительно, пептид, заякоривающий в митохондриях, может являться пептидом, который может локализоваться на митохондриальной внешней мембране. Таким образом, чужеродный белок может быть связан с внешней мембраной митохондрий с помощью пептида, заякоривающего в митохондриях. Пептид, заякоривающий в митохондриях, может являться пептидом, содержащим N-концевую область или C-концевую область белка, присутствующего в митохондриальном мембранном белке, и N-концевую область или C-концевую область белка, присутствующего во внешней мембране митохондрий, белок может локализоваться на внешней мембране митохондрий. В этом случае заякоривающий пептид может дополнительно содержать митохондриальную сигнальную последовательность.
Один из вариантов осуществления белка, присутствующего в митохондриальной мембране, может быть выбран из группы, состоящей из TOM20, TOM70, OM45, TOM5, TOM6, TOM7, TOM22, Fis1, Bcl-2, Bcl-x и VAMP1B. В частности, если пептид, заякоривающий в митохондриях, получают из любого пептида, выбранного из группы, состоящей из TOM20, TOM70 и OM45, он может содержать N-концевую область TOM20, TOM70 или OM45. Одним из вариантов осуществления пептида, заякоривающего в митохондриях, может являться TOM70, полученный из дрожжей и приведенный в SEQ ID NO: 75, или TOM70, полученный из человека и приведенный в SEQ ID NO: 76. Другим вариантом осуществления может являться TOM20, полученный из дрожжей и приведенный в SEQ ID NO: 77, или TOM20, полученный из человека и приведенный в SEQ ID NO: 78. Другим вариантом осуществления может являться OM45, полученный из дрожжей и приведенный в SEQ ID NO: 79.
Кроме того, если пептид, заякоривающий в митохондриях, получают из любого пептида, выбранного из группы, состоящей из TOM5, TOM6, TOM7, TOM22, Fis1, Bcl-2, Bcl-x и VAMP1B, он может содержать C-концевую область любого пептида, выбранного из группы, состоящей из TOM5, TOM6, TOM7, TOM22, Fis1, Bcl-2, Bcl-x и VAMP1B. Одним из вариантов осуществления пептида, заякоривающего в митохондриях, может являться TOM5, полученный из дрожжей и приведенный в SEQ ID NO: 80, или TOM5, полученный из человека и приведенный в SEQ ID NO: 81. Другим вариантом осуществления может являться TOM7, полученный из дрожжей и приведенный в SEQ ID NO: 82, или TOM7, полученный из человека и приведенный в SEQ ID NO: 83. Другим вариантом осуществления может являться TOM22, полученный из дрожжей и приведенный в SEQ ID NO: 84, или TOM22, полученный из человека и приведенный в SEQ ID NO: 85. Другим вариантом осуществления может являться Fis1, полученный из дрожжей и приведенный в SEQ ID NO: 86, или Fis1, полученный из человека и приведенный в SEQ ID NO: 87. Другим вариантом осуществления может являться Bcl-2 альфа, полученный из человека и приведенный в SEQ ID NO: 88. Другим вариантом осуществления может являться VAMP1, полученный из дрожжей и приведенный в SEQ ID NO: 89, или VAMP1, полученный из человека и приведенный в SEQ ID NO: 90.
В этом случае желаемый белок, способный функционировать внутри и вне клетки и включенный в чужеродный белок, может являться любым белком, выбранным из группы, состоящей из активного белка, проявляющего активность в клетке, белка, присутствующего в клетке, и белка, имеющего способность связываться с лигандом или рецептором, присутствующим в мембране клетки.
Вариант осуществления активного белка или белка, присутствующего в клетке, может являться любым белком, выбранным из группы, состоящей из p53, гранзима B, Bax, Bak, PDCD5, E2F, AP-1 (Jun/Fos), EGR-1, белка ретинобластомы (RB), гомолога фосфатазы и тензина (PTEN), E-кадгерина, нейрофибромина-2 (NF-2), поли-АДФ-рибоза-синтазы 1 (PARP-1), BRCA-1, BRCA-2, белка аденоматозного полипоза толстой кишки (APC), фактора, ассоциированного с рецептором фактора некроза опухоли (TRAF), Ингибиторного белка RAF-киназы (RKIP), p16, KLF-10, LKB1, LHX6, C-RASSF, DKK-3PD1, Oct3/4, Sox2, Klf4 и c-Myc. Если желаемый белок выбран из указанной выше группы, его можно связывать с заякоривающим пептидом, содержащим N-концевую область TOM20, TOM70 или OM45.
Такой слитый белок можно связывать в следующем порядке:
N-конец-заякоривающий пептид, содержащий N-концевую область TOM20, TOM70 или OM45-желаемый белок-C-конец.
Кроме того, чужеродный белок может дополнительно содержать аминокислотную последовательность, распознаваемую протеолитическим ферментом в эукариотических клетках, или убиквитин или его фрагмент между пептидом, заякоривающим в митохондриях, и желаемым белком. Термин "протеолитический фермент" в эукариотических клетках относится к ферменту, приводящему к деградации белка, присутствующего в эукариотических клетках. В этом случае, т.к. чужеродный белок содержит аминокислотную последовательность, распознаваемую ферментом, приводящим к деградации белка, чужеродный белок, связанный с митохондриальной внешней мембраной, может разделяться в клетке на заякоривающий пептид и желаемый белок.
В этом случае фрагмент убиквитина может содержать C-концевую аминокислотную последовательность Gly-Gly SEQ ID NO: 71 и может содержать от 3 до 75 аминокислот, расположенных последовательно от C-конца. Кроме того, чужеродный белок может дополнительно содержать линкер между желаемым белком и убиквитином или его фрагментом. В этом случае линкер может состоять, в качестве неограничивающих примеров из от 1 до 150 аминокислот, или от 10 до 100 аминокислот, или от 20 до 50 аминокислот. Линкер может состоять из аминокислот, соответствующим образом выбранных из 20 аминокислот, предпочтительно, может состоять из глицина и/или серина. Один из вариантов осуществления линкера может состоять из от 5 до 50 аминокислот, состоящих из глицина и серина. Один из вариантов осуществления линкера может представлять собой (G4S)n, в котором n является целым числом от 1 до 10, и n может составлять 1, 2, 3, 4, 5, 6, 7, 8, 9 или 10.
Кроме того, белок, имеющий способность связываться с лигандом или рецептором, присутствующим в мембране клетки, может являться лигандом или рецептором, присутствующим на поверхности опухолевой клетки. В этом случае лиганд или рецептор, присутствующий на поверхности опухолевой клетки, может являться, в качестве неограничивающих примеров, любым лигандом или рецептором, выбранным из группы, состоящей из CD19, CD20, меланома-ассоциированного антигена E(MAGE), NY-ESO-1, карциноэмбрионального антигена (CEA), муцина 1, ассоциированного с поверхностью клетки (MUC-1), простатической кислой фосфатазы (PAP), простат-специфического антигена (PSA), сурвивина, связанного с тирозиназой белка 1 (tyrp1), связанного с тирозиназой белка 1 (tyrp2), гена брахиурии, мезотелина, рецептора эпидермального фактора роста (EGFR), рецептора эпидермального фактора роста 2 человека (HER-2), ERBB2, белка опухоли Вильмса (WT1), FAP, EpCAM, PD-L1, ACPP, CPT1A, ИФНΓ, CD274, FOLR1, EPCAM, ICAM2, NCAM1, LRRC4, UNC5H2 LILRB2, CEACAM, нектина-3 и их комбинации.
Кроме того, белок, имеющий способность связываться с лигандом или рецептором, присутствующим в мембране клетки, может являться антителом или его фрагментом, связывающимся с любым белком, выбранным из указанной выше группы. В частности, термин "фрагмент антитела" относится к фрагменту, имеющему ту же определяющую комплементарность область (CDR), что и антитело. В частности, он может являться Fab, scFv, F(ab')2 или их комбинацией.
В этом случае желаемый белок может быть связан с заякоривающим пептидом, содержащим C-концевую область любого белка, выбранного из группы, состоящей из TOM5, TOM6, TOM7, TOM22, Fis1, Bcl-2, Bcl-x и VAMP1B, и чужеродный белок может быть связан в следующем порядке:
N-конец-желаемый белок-заякоривающий пептид, содержащий C-концевую область любого белка, выбранного из группы, состоящей из TOM5, TOM6, TOM7, TOM22, Fis1, Bcl-2, Bcl-x и VAMP1B-C конец.
Кроме того, чужеродный белок может дополнительно содержать линкер между желаемым белком и C-концевой областью любого белка, выбранного из группы, состоящей из TOM5, TOM6, TOM7, TOM22, Fis1, Bcl-2, Bcl-x и VAMP1B. В этом случае линкер является таким, как описано выше. В этом случае желаемый белок, активный белок, белок, присутствующий в клетке, и белок, имеющий способность связываться с лигандом или рецептором, присутствующим в мембране клетки и т.п., являются такими, как описано выше.
В одном из вариантов осуществления желаемого белка антитело или его фрагмент против конкретной клетки может находиться в форме, связанной с заякоривающим пептидом, содержащим C-концевую область любого белка, выбранного из группы, состоящей из TOM5, TOM6, TOM7, TOM22, Fis1, Bcl-2, Bcl-x и VAMP1B. Модифицированные митохондрии, с которыми связан желаемый белок, легко можно вводить в конкретную мишень таким образом, что митохондрии могут эффективно проникать в конкретную клетку.
Один из вариантов осуществления модифицированных митохондрий может находиться в форме, с которой связаны один или более желаемых белков. В частности, они могут находиться в форме, с которой связан желаемый белок, содержащий p53, и желаемый белок, содержащий антитело против HER-2 или его фрагмент. Такие модифицированные митохондрии могут подвергаться эффективной доставке в злокачественные клетки, экспрессирующие HER-2. Кроме того, злокачественные клетки могут эффективно подвергаться уничтожению посредством p53, связанного с модифицированными митохондриями.
В зависимости от предназначения модифицированных митохондрий, можно конструировать желаемый белок, содержащий один или более активных белков, и можно позволять ему связываться с митохондриями. Кроме того, желаемый белок, направленно воздействующий на клетку, можно конструировать различными способами в зависимости от клетки-мишени.
В другом аспекте настоящее изобретение относится к фармацевтической композиции, содержащей описанные выше модифицированные митохондрии в качестве активного ингредиента. В этом случае фармацевтическую композицию можно использовать для профилактики или лечения злокачественного новообразования. В этом случае злокачественное новообразование может являться злокачественным новообразованием, выбранным из группы, состоящей из рака желудка, рака печени, рака легких, колоректального рака, рака молочной железы, рака предстательной железы, рака яичников, рака поджелудочной железы, рака шейки матки, рака щитовидной железы, рака гортани, острого миелоидного лейкоза, опухоли головного мозга, нейробластомы, ретинобластомы, рака головы и шеи, рака слюнных желез и лимфомы.
В частности, если активный белок уничтожает опухолевые клетки, подобно p53, или если белок, ингибирующий пролиферацию, связан с митохондриями, модифицированные митохондрии, с которыми связан p53, можно использовать в качестве противоопухолевого средства. Кроме того, если белок, такой как RKIP, способный ингибировать метастазирование злокачественных клеток, связан с митохондриями, модифицированные митохондрии, с которыми связан RKIP, можно использовать в качестве ингибитора метастазирования опухоли. Если с митохондриями связан любой белок, выбранный из группы, состоящей из гранзима B, Bax, Bak, PDCD5, E2F, AP-1 (Jun/Fos), EGR-1, белка ретинобластомы (RB), гомолога фосфатазы и тензина (PTEN), E-кадгерина, нейрофибромина-2(NF-2), поли-АДФ-рибоза-синтазы 1 (PARP-1), BRCA-1, BRCA-2, белка аденоматозного полипоза толстой кишки (APC), фактора, ассоциированного с рецептором фактора некроза опухоли (TRAF), p16, KLF-10, LKB1, LHX6, C-RASSF, DKK-3PD1 и их комбинации, являющихся белками, ингибирующими пролиферацию злокачественных клеток, или контролирующими реакцию фосфорилирования в злокачественных клеток, или ингибирующими метастазирование злокачественных клеток, модифицированные митохондрии, с которыми связан активный белок, можно использовать в качестве противоопухолевого средства.
Кроме того, в случае фармацевтической композиции митохондрии можно включать в концентрации, в качестве неограничивающих примеров, от 0,1 мкг/мл до 500 мкг/мл, от 0,2 мкг/мл до 450 мкг/мл или от 0,5 мкг/мл до 400 мкг/мл. Включение митохондрий в указанном диапазоне может облегчать корректировку дозы митохондрий после введения и может повышать степень улучшения симптомов заболевания у пациента. В этом случае дозу митохондрий можно определять посредством количественного анализа митохондрий посредством подсчета мембранного белка в выделенных митохондриях. В частности, выделенные митохондрии можно количественно анализировать посредством анализа белков Брэдфорда (статья James D. McCully (J Vis Exp. 2014; (91): 51682)).
Кроме того, в случае фармацевтической композиции активный белок, связывающийся с митохондриями, можно включать в концентрации, в качестве неограничивающих примеров, от 0,1 мкг/мл до 500 мкг/мл, от 0,2 мкг/мл до 450 мкг/мл или от 0,5 мкг/мл до 400 мкг/мл. Включение активного белка в указанном выше диапазоне может облегчать корректировку дозы активного белка после введения и может повышать степень улучшения симптомов заболевания у пациента.
Кроме того, в случае фармацевтической композиции белок для таргетинга, способный доставлять митохондрии в конкретную клетку, можно включать в концентрации, в качестве неограничивающих примеров, от 0,1 мкг/мл до 500 мкг/мл, от 0,2 мкг/мл до 450 мкг/мл, от 0,5 мкг/мл до 400 мкг/мл. Включение белка для таргетинга в указанном выше диапазоне может облегчать корректировку дозы белка для таргетинга после введения и может повышать степень улучшения симптомов заболевания у пациента.
В частности, фармацевтическую композицию по настоящему изобретению можно вводить с митохондриями в количестве, в качестве неограничивающих примеров, 0,01-5 мг/кг, 0,1-4 мг/кг или 0,25-2,5 мг/кг за один раз в пересчете на массу тела индивидуума, которому будут их вводить. Т.е. наиболее предпочтительным в терминах активности клетки является введение фармацевтической композиции таким, чтобы количество модифицированных митохондрий попадало в указанный выше диапазон в пересчет на массу тела индивидуума, имеющего злокачественные ткани. Кроме того, фармацевтическую композицию можно вводить 1-10 раз, 3-8 раз или 5-6 раз и, предпочтительно, 5 раз. В этом случае интервал между введениями может составлять 1-7 дней или 2-5 дней и, предпочтительно, 3 дня.
Кроме того, фармацевтическую композицию по настоящему изобретению можно вводить человеку или другому млекопитающему, склонному к развитию злокачественного новообразования или страдающего злокачественным новообразованием. Кроме того, фармацевтическая композиция может являться инъецируемым препаратом, который можно вводить внутривенно, или инъецируемым препаратом, который можно вводить местно, и, предпочтительно, может являться препаратом для инъекций.
Таким образом, фармацевтическую композицию по настоящему изобретению можно получать в виде физически или химически высокостабильного препарата посредством коррекции pH композиции с помощью буферного раствора, такого как водный раствор кислоты или фосфат, который можно использовать в инъецируемом препарате, для обеспечения стабильности продукта во время распространения инъецируемых препаратов.
В частности, фармацевтическая композиция по настоящему изобретению может содержать воду для инъекций. Вода для инъекций представляет собой дистиллированную воду, подготовленную для растворения твердого инъецируемого препарата или разведения водорастворимого инъецируемого препарата, и может являться глюкозой для инъекций, ксилитом для инъекций, D-маннитом для инъекций, фруктозой для инъекций, физиологическим раствором, декстраном 40 для инъекций, декстраном 70 для инъекций, аминокислотами для инъекций, раствором Рингера, лактатом Рингера, фосфатным буферным раствором, имеющим pH от 3,5 до 7,5, буферным раствором дигидрофосфата-цитрата натрия или т.п.
Кроме того, фармацевтическая композиция по настоящему изобретению может включать стабилизатор или средство для растворения. Например, стабилизатор может являться пиросульфитом натрия или этилендиаминтетрауксусной кислотой, и средство для растворения может являться соляной кислотой, уксусной кислотой, гидроксидом натрия, гидрокарбонатом натрия, карбонатом натрия или гидроксидом калия.
Кроме того, настоящее изобретение может относиться к способу профилактики или лечения злокачественного новообразования, включающему введение индивидууму указанной выше фармацевтической композиции. В настоящем описании индивидуум может являться млекопитающим, и предпочтительно - человеком.
В одном из аспектов настоящее изобретение относится к способу получения модифицированных митохондрий, включающему стадию смешивания выделенных митохондрий с желаемым белком, содержащим активный белок, и/или желаемым белком, содержащим белок для таргетинга.
В этом случае желаемый белок и митохондрии можно смешивать в подходящем соотношении. Например, желаемый белок:митохондрии можно смешивать в соотношении от 1:100 до 100:1 в пересчете на массу. В частности, их можно смешивать в соотношении 1:10, 1:5, 1:4, 1:3, 1:2 или 1:1. Кроме того, соотношение может составлять 10:1, 5:1, 4:1, 3:1 или 2:1.
В другом аспекте настоящее изобретение относится к способу получения модифицированных митохондрий из трансформированных клеток посредством инъецирования полинуклеотида, кодирующего описанный выше желаемый белок, в эукариотические клетки. В частности, настоящее изобретение относится к способу получения описанного выше слитого белка, включающему стадию трансформации прокариотических клеток или эукариотических клеток с использованием описанного выше полинуклеотида без фермента деградации убиквитина или протеолитического фермента в эукариотических клетках; и стадию получения слитого белка. Этот способ получения является подходящим, если желаемый белок не содержит аминокислотную последовательность, распознаваемую протеолитическим ферментом в эукариотических клетках, или убиквитин или его фрагмент.
В другом аспекте настоящего изобретения желаемый белок можно получать с использованием прокариотической клетки или экстракта прокариотических клеток. Кроме того, настоящее изобретение относится к способу получения модифицированных митохондрий с использованием эукариотических клеток без фермента деградации убиквитина или протеолитического фермента или экстракта эукариотических клеток.
В другом аспекте настоящее изобретение относится к применению митохондрий в качестве средств доставки чужеродного белка. В частности, модифицированные митохондрии можно использовать в качестве средств внутриклеточной и внеклеточной доставки чужеродного белка, содержащего желаемый белок, способный функционировать внутри и вне клетки. Митохондрии можно эффективно встраивать в клетки, и в этом случае чужеродный белок, который желательно доставлять в клетки, можно эффективно доставлять в клетки. В этом случае митохондрии можно использовать в качестве эффективной системы доставки белка. Желаемый белок является таким, как описано выше.
Другой аспект настоящего изобретения относится к слитому белку, содержащему пептид, заякоривающий в митохондриальной внешней мембране, и желаемый белок. В этом случае желаемый белок является таким, как описано выше.
В рамках изобретения термин "пептид, заякоривающий в митохондриальной внешней мембране" может означать N-конец или C-конец белка, присутствующего во внешней мембране митохондрий. Пептид, заякоривающий в митохондриальной внешней мембране, может иметь аминокислотную последовательность, конкретно локализованную во внешней мембране митохондрий. В этом случае пептид, заякоривающий в митохондриальной внешней мембране, позволяет прикреплять слитый белок, представленный в настоящем описании, к внешней мембране митохондрий. В этом случае пептид, заякоривающий в митохондриальной внешней мембране, можно использовать в том же смысле, что и пептид для таргетинга митохондриальной внешней мембраны.
Кроме того, пептид, заякоривающий в митохондриальной внешней мембране, предотвращает проникновение слитого белка, представленного в настоящем описании, внутрь митохондрий. Комплекс TOM (транслоказы внешней мембраны), присутствующий в митохондриальной внешней мембране, имеет митохондриальную последовательность-мишень и отдельный домен заякоривания во внешней мембране на амино-конце, и большая часть карбокси-конца может иметь структуру, экспонируемую в направлении цитоплазмы (фигура 81a). Комплекс TOM (транслоказы внешней мембраны), присутствующий в митохондриальной внешней мембране, имеет митохондриальную последовательность-мишень и отдельный домен заякоривания во внешней мембране на карбокси-конце, и большая часть амино-конца также может иметь структуру, экспонируемую в направлении цитоплазмы (фигура 81b). Кроме того, белок, присутствующий во внешней мембране митохондрий может быть выбран из белков, присутствующих в митохондриях, находящихся в эукариотической клетке. Например, он может быть выбран из белков, присутствующих в митохондриальной внешней мембране, находящейся в дрожжах, клетках животных или клетках человека.
В этом случае вариант осуществления белка, присутствующего в митохондриальной внешней мембране, может являться любым белком, выбранным из группы, состоящей из TOM20, TOM70, OM45, TOM5, TOM6, TOM7, TOM22, Fis1, Bcl-2, Bcl-x и VAMP1B или его фрагмента. В этом случае пептид, заякоривающий в митохондриальной внешней мембране, может являться фрагментом любого белка, выбранного из группы, состоящей из TOM20, TOM70, OM45, TOM5, TOM6, TOM7, TOM22, Fis1, Bcl-2, Bcl-x и VAMP1B. В этом случае пептид, заякоривающий в митохондриальной внешней мембране, может являться C-концевым или N-концевым полипептидом TOM20, TOM70, OM45, TOM5, TOM6, TOM7, TOM22, Fis1, Bcl-2, Bcl-x и VAMP1B, локализованным в митохондриальной внешней мембране.
В частности, если пептид, заякоривающий в митохондриальной внешней мембране, слит с N-концом желаемого белка, пептид, заякоривающий в митохондриальной внешней мембране, может содержать концевую последовательность белка, выбранного из группы, состоящей из TOM20, TOM70 и OM45. Предпочтительно, она может являться N-концевой последовательностью белка, выбранного из группы, состоящей из TOM20, TOM70 и OM45. Вариант осуществления пептида, заякоривающего в митохондриальной внешней мембране, является таким, как описано выше.
Кроме того, если пептид, заякоривающий в митохондриальной внешней мембране, слит с C-концом желаемого белка, белок для таргетинга внешней мембраны может содержать концевую последовательность белка, выбранного из группы, состоящей из TOM5, TOM6, TOM7, TOM22, Fis1, Bcl-2, Bcl-X и VAMP1B. Предпочтительно, она может являться C-концевой последовательностью белка, выбранного из группы, состоящей из TOM5, TOM6, TOM7, TOM22, Fis1, Bcl-2, Bcl-X и VAMP1B. Вариант осуществления пептида, заякоривающего в митохондриальной внешней мембране, является таким, как описано выше.
В рамках изобретения термин "активный белок" может представлять собой белок, проявляющий физиологическую активность. Одним из вариантов осуществления такого активного белка может являться белок, имеющий сниженную функцию, или модифицированный белок, присутствующий в поврежденных злокачественных клетках. Одним из вариантов осуществления активного белка может являться белок, повышающий активность клеток. Вариант осуществления такого активного белка является таким, как описано выше.
Слитый белок может являться белком, с которым связаны белок для таргетинга митохондриальной внешней мембраны и желаемый белок, с N-конца до C-конца. В этом случае он может дополнительно содержать убиквитин или его фрагмент, имеющий специфический участок расщепления убиквитиновых протеаз (глицин-глицин) между белком для таргетинга митохондриальной внешней мембраны и желаемым белком. В этом случае для облегчения расщепления убиквитиновой протеазой он может дополнительно содержать линкер, содержащий гидрофильные и полярные аминокислоты, серин, глицин и треонин, между белком для таргетинга митохондриальной внешней мембраны и белком убиквитином.
В рамках изобретения термин "убиквитин" относится к белку, участвующему в протеолитическом процессе, также обозначаемому как UB. Одним из вариантов осуществления убиквитина может являться убиквитин, присутствующий в организме человека, или убиквитин, присутствующий в дрожжах. Убиквитин, присутствующий в организме человека, состоит из 76 аминокислот. В этом случае убиквитин можно использовать в зрелой форме. В рамках изобретения термин "зрелая форма" может относиться к белку в форме, из которой удален сигнальный пептид.
Кроме того, фермент, обозначаемый как убиквитиновая протеаза или UBP (убиквитин-специфическая протеаза), от природы присутствует в эукариотических клетках и может индуцировать природную диссоциацию желаемого белка посредством расщепления C-концевого аминокислотного глицин-глицинового участка убиквитина в клетке.
В этом случае фрагмент убиквитина может содержать аминокислоты Gly-Gly C-конца убиквитина и может содержать от 3 до 75 последовательных аминокислот с C-конца. В частности, вариантом осуществления фрагмента убиквитина может являться Arg-Gly-Gly, Leu-Arg-Gly-Gly, Arg-Leu-Arg-Gly-Gly или Leu-Arg-Leu-Arg-Gly-Gly. Кроме того, фрагмент убиквитина может иметь аминокислотную последовательность SEQ ID NO: 71.
Слитый белок, содержащий белок для таргетинга митохондриальной внешней мембраны и желаемый белок, можно обозначать как слитый белок, модифицирующий активность митохондрий. Такой слитый белок может иметь любую из следующих структур:
<Структурная формула 1>
N-конец-пептид, заякоривающий в митохондриальной внешней мембране-желаемый белок-C-конец
<Структурная формула 2>
N-конец-пептид, заякоривающий в митохондриальной внешней мембране-убиквитин или его фрагмент-желаемый белок-C-конец
<Структурная формула 3>
N-конец-белок для таргетинга митохондриальной внешней мембраны-линкер 1-убиквитин или его фрагмент-желаемый белок-C-конец
<Структурная формула 4>
N-конец-пептид, заякоривающий в митохондриальной внешней мембране-убиквитин или его фрагмент-линкер 2-желаемый белок-C-конец
<Структурная формула 5>
N-конец-пептид, заякоривающий в митохондриальной внешней мембране-линкер 1-убиквитин или его фрагмент-линкер 2-желаемый белок-C-конец
В указанных выше структурных формулах 1-5, пептид, заякоривающий во внешней мембране, может являться концевой последовательностью белка, выбранного из группы, состоящей из TOM20, TOM70 и OM45, и желаемый белок может являться любым белком, выбранным из группы, состоящей из p53, гранзима B, Bax, Bak, PDCD5, E2F, AP-1 (Jun/Fos), EGR-1, белка ретинобластомы (RB), гомолога фосфатазы и тензина (PTEN), E-кадгерина, нейрофибромина-2 (NF-2), поли-АДФ-рибоза-синтазы 1 (PARP-1), BRCA-1, BRCA-2, белка аденоматозного полипоза толстой кишки (APC), фактора, ассоциированного с рецептором фактора некроза опухоли (TRAF), ингибиторного белка RAF-киназы (RKIP), p16, KLF-10, LKB1, LHX6, C-RASSF и DKK-3PD1.
В этом случае линкер 1 или 2 может являться полипептидом, состоящим из от 1 до 100, от 1 до 80, от 1 до 50 или от 1 до 30 аминокислот, соответственно, и, предпочтительно, может являться полипептидом, состоящим из от 1 до 30 аминокислот, состоящих из серина, глицина или треонина в отдельности или в комбинации. Кроме того, линкер 1 или 2 может являться полипептидом, состоящим из от 5 до 15 аминокислот, соответственно, и, предпочтительно, может являться полипептидом, состоящим из от 5 до 15 аминокислот, состоящих из серина, глицина или треонина в отдельности или в комбинации. Одним из вариантов осуществления линкера может являться (GGGGS)3 (SEQ ID NO: 70).
<Структурная формула 6>
N-конец-желаемый белок-пептид, заякоривающий в митохондриальной внешней мембране-C-конец
<Структурная формула 7>
N-конец-желаемый белок-убиквитин или его фрагмент-пептид, заякоривающий в митохондриальной внешней мембране-C-конец
<Структурная формула 8>
N-конец-желаемый белок-линкер 1-убиквитин или его фрагмент-пептид, заякоривающий в митохондриальной внешней мембране-C-конец
<Структурная формула 9>
N-конец-желаемый белок-убиквитин или его фрагмент-линкер 2-пептид, заякоривающий в митохондриальной внешней мембране-C-конец
<Структурная формула 10>
N-конец-желаемый белок-линкер 1-убиквитин или его фрагмент-линкер 2-пептид для таргетинга митохондриальной внешней мембраны-C-конец
В указанных выше структурных формулах 6-10 пептид, заякоривающий во внешней мембране, может являться концевой последовательностью белка, выбранного из группы, состоящей из TOM5, TOM6, TOM7, TOM22, Fis1, Bcl-2, Bcl-X и VAMP1B, и желаемый белок может являться любым белком, выбранным из группы, состоящей из p53, гранзима B, Bax, Bak, PDCD5, E2F, AP-1 (Jun/Fos), EGR-1, белка ретинобластомы (RB), гомолога фосфатазы и тензина (PTEN), E-кадгерина, нейрофибромина-2(NF-2), поли-АДФ-рибоза-синтазы 1 (PARP-1), BRCA-1, BRCA-2, белка аденоматозного полипоза толстой кишки (APC), фактора, ассоциированного с рецептором фактора некроза опухоли (TRAF), ингибиторного белка RAF-киназы (RKIP), p16, KLF-10, LKB1, LHX6, C-RASSF, DKK-3PD1, Oct3/4, Sox2, Klf4 и c-Myc. В этом случае линкер 1 или 2 является таким, как описано выше.
В одном из аспектов настоящее изобретение относится к полинуклеотиду, кодирующему слитый белок, содержащий пептид, заякоривающий в митохондриальной внешней мембране, и желаемый белок.
Кроме того, в одном из аспектов настоящее изобретение относится к вектору, нагруженному полинуклеотидом, кодирующим слитый белок, содержащий желаемый белок.
Кроме того, в одном из аспектов настоящее изобретение относится к клетке-хозяину, в которую встраивают вектор, нагруженный полинуклеотидом, кодирующим слитый белок, содержащий желаемый белок.
В одном из аспектов настоящее изобретение относится к слитому белку, содержащему белок для таргетинга мишени и белок для таргетинга митохондриальной внешней мембраны.
В этом случае белок для таргетинга мишени и пептид, заякоривающий в митохондриальной внешней мембране, могут быть связаны от N-конца к C-концу. В этом случае пептид, заякоривающий в митохондриальной внешней мембране, может являться любым пептидом, выбранным из группы, состоящей из TOM20, TOM70, OM45, TOM5, TOM6, TOM7, TOM22, Fis1, Bcl-2, Bcl-x и VAMP1B.
В рамках изобретения термин "мишень" относится к месту, в которое необходимо доставить модифицированные митохондрии. Одним из вариантов осуществления мишени может являться злокачественная клетка. В частности, одним из вариантов осуществления мишени может являться биомаркер, находящийся на поверхности злокачественных клеток. В частности, мишень может являться опухолеспецифическим антигеном (TAA). В этом случае опухолеспецифический антиген может являться любым антигеном, выбранным из группы, состоящей из CD19, CD20, меланома-ассоциированного антигена E (MAGE), NY-ESO-1, карциноэмбрионального антигена (CEA), муцина 1, ассоциированного с поверхностью клетки (MUC-1), простатической кислой фосфатазы (PAP), простат-специфического антигена (PSA), сурвивина, тирозиназа-связанного белка 1 (tyrp1), тирозиназа-связанного белка 1 (tyrp2), гена брахиурии, мезотелина, рецептора эпидермального фактора роста (EGFR), рецептора эпидермального фактора роста 2 человека (HER-2), ERBB2, белка опухоли Вильмса (WT1), FAP, EpCAM, PD-L1, ACPP, CPT1A, ИФНΓ, CD274, FOLR1, EPCAM, ICAM2, NCAM1, LRRC4, UNC5H2 LILRB2, CEACAM, нектина-3 и их комбинации.
В рамках изобретения термин "белок для таргетинга мишени" может означать белковую последовательность, способную связываться с описанной выше мишенью. В этом случае одним из вариантов осуществления белка для таргетинга мишени может являться белок, связывающийся с биомаркером, находящимся на поверхности злокачественных клеток. В этом случае вариантом осуществления биомаркера, находящегося на поверхности злокачественных клеток, может являться, в качестве неограничивающих примеров, ICAM2, NCAM1, LRRC4, UNC5H2, LILRB2, CEACAM или нектин-3. В этом случае белок для таргетинга мишени можно включать в описанный выше чужеродный белок.
Одним из вариантов осуществления белка для таргетинга мишени может являться антитело или его фрагмент. В частности, он может являться антителом или его фрагментом, специфически связывающимся с опухолеспецифическим антигеном. Кроме того, фрагмент антитела может являться любым фрагментом, выбранным из группы, состоящей из Fab, Fab', scFv и F(ab)2.
Одним из вариантов осуществления белка для таргетинга мишени может являться scFvHER, способный связываться с рецептором эпидермального фактора роста. Другим вариантом осуществления может являться scFvMEL, с помощью которого можно осуществлять таргетинг меланомы. Другим вариантом осуществления может являться scFvPD-L1, способный связываться с PD-L1, гиперэкспрессированным на поверхности злокачественных клеток. Другим вариантом осуществления может являться PD-1, способный связываться с PDL-1, гиперэкспрессированным на поверхности злокачественных клеток.
Один из аспектов настоящего изобретения может дополнительно содержать убиквитин или его фрагмент между белком для таргетинга мишени и белком для таргетинга митохондриальной внешней мембраны. Слитый белок, содержащий белок для таргетинга митохондриальной мишени и желаемый белок, можно обозначать как слитый белок, модифицирующий активность митохондрий. Такой слитый белок может иметь любую из следующих структур:
<Структурная формула 11>
N-конец-белок для таргетинга мишени-пептид, заякоривающий в митохондриальной внешней мембране-C-конец
<Структурная формула 12>
N-конец-белок для таргетинга мишени-убиквитин или его фрагмент-пептид, заякоривающий в митохондриальной внешней мембране-C-конец
<Структурная формула 13>
N-конец-белок для таргетинга мишени-линкер 1-убиквитин или его фрагмент-пептид, заякоривающий в митохондриальной внешней мембране-C-конец
<Структурная формула 14>
N-конец-белок для таргетинга мишени-убиквитин или его фрагмент-линкер 2-пептид, заякоривающий в митохондриальной внешней мембране-C-конец
<Структурная формула 15>
N-конец-белок для таргетинга мишени-линкер 1-убиквитин или его фрагмент-линкер 2-пептид, заякоривающий в митохондриальной внешней мембране-C-конец
В указанных выше структурных формулах 11-15 пептид, заякоривающий во внешней мембране, может являться концевой последовательностью белка, выбранной из группы, состоящей из TOM5, TOM6, TOM7, TOM22, Fis1, Bcl-2, Bcl-X и VAMP1B, и белок для таргетинга мишени может являться любым белком, выбранным из группы, состоящей из опухолеассоциированных антигенов, CD19, CD20, меланома-ассоциированного антигена E (MAGE), NY-ESO-1, карциноэмбрионального антигена (CEA), муцина 1, ассоциированного с поверхностью клетки (MUC-1), простатической кислой фосфатазы (PAP), простат-специфического антигена (PSA), сурвивина, тирозиназа-связанного белка 1 (tyrp1), тирозиназа-связанного белка 1 (tyrp2), гена брахиурии, мезотелина, рецептора эпидермального фактора роста(EGFR), рецептора эпидермального фактора роста 2 человека (HER-2), ERBB2, белка опухоли Вильмса (WT1), FAP, EpCAM, PD-L1, ACPP, CPT1A, ИФНΓ, CD274, FOLR1, EPCAM, ICAM2, NCAM1, LRRC4, UNC5H2 LILRB2, CEACAM, нектина-3 и их комбинации. Кроме того, белок для таргетинга мишени может являться антителом, специфически связывающимся с опухолеассоциированным антигеном или его фрагментом. В этом случае линкер 1 или 2 и аминокислотная последовательность, распознаваемая протеолитическим ферментом, являются такими, как описано выше.
<Структурная формула 16>
N-конец-пептид, заякоривающий в митохондриальной внешней мембране-белок для таргетинга мишени-C-конец
<Структурная формула 17>
N-конец-пептид, заякоривающий в митохондриальной внешней мембране-убиквитин или его фрагмент-белок для таргетинга мишени-C-конец
<Структурная формула 18>
N-конец-пептид, заякоривающий в митохондриальной внешней мембране-линкер 1-убиквитин или его фрагмент-белок для таргетинга мишени-C-конец
<Структурная формула 19>
N-конец-пептид, заякоривающий в митохондриальной внешней мембране-убиквитин или его фрагмент-линкер 2-белок для таргетинга мишени-C-конец
<Структурная формула 20>
N-конец-пептид, заякоривающий в митохондриальной внешней мембране-линкер 1-убиквитин или его фрагмент-линкер 2-белок для таргетинга мишени-C-конец
В указанных выше структурных формулах 16-20 пептид, заякоривающий во внешней мембране, может являться любым пептидом, выбранным из группы, состоящей из TOM20, TOM70 и OM45. Кроме того, белок для таргетинга мишени, убиквитин или его фрагмент и линкер 1 или 2 являются такими, как описано выше.
В одном из аспектов настоящее изобретение относится к полинуклеотиду, кодирующему слитый белок, содержащий белок для таргетинга мишени.
Кроме того, в одном из аспектов настоящее изобретение относится к вектору, нагруженному полинуклеотидом, кодирующим слитый белок, содержащий белок для таргетинга мишени.
Кроме того, в одном из аспектов настоящее изобретение относится к клетке-хозяину, в которую встраивают вектор, нагруженный полинуклеотидом, кодирующим слитый белок, содержащий белок для таргетинга мишени. Клетка-хозяин может являться прокариотической клеткой или эукариотической клеткой. В этом случае, предпочтительно, эукариотическая клетка может представлять собой штамм, из которого удален фермент, приводящий к деградации убиквитина.
Кроме того, в одном из аспектов настоящее изобретение относится к способу получения модифицированных митохондрий из трансформированных клеток посредством инъецирования полинуклеотида, кодирующего слитый белок, в эукариотические клетки.
СПОСОБ ОСУЩЕСТВЛЕНИЯ ИЗОБРЕТЕНИЯ
Далее в настоящем описании для облегчения понимания настоящего изобретения будет представлен предпочтительный вариант осуществления. Однако следующие примеры представлены исключительно для облегчения понимания настоящего изобретения, и настоящее изобретение ими не ограничено.
I. Получение слитого белка, содержащего пептид, заякоривающий в митохондриальной внешней мембране, линкер, убиквитин и желаемый белок
Пример 1. Получение слитого белка, содержащего p53
Пример 1.1. Амплификация гена p53
Для экспрессии p53 человека в рекомбинантном белке выделяли тотальную РНК из эпителиальных клеток человека и из нее синтезировали кДНК. В частности, дермальные фибробласты человека культивировали в среде с 10% сыворотки в условиях 5% диоксида углерода и 37°C (1×106 клеток). Затем удаляли культуральный раствор, дважды промывали клетки посредством добавления к ним буферного раствора PBS и напрямую добавляли 0,5 мл экстракта РНК (реагент тризол, Thermo Fisher Scientific). Смесь, к которой добавляли экстракт РНК, оставляли при температуре окружающей среды на 10 минут, а затем добавляли 0,1 мл хлороформа и перемешивали в течение 15 секунд, а затем центрифугировали при приблизительно 12000×g в течение 10 минут. Затем отбирали отделенный супернатант и добавляли тот же объем изопропилового спирта и снова центрифугировали при 12000×g в течение 10 минут. Затем удаляли жидкость и однократно промывали 75% этанолом и сушили РНК при температуре окружающей среды.
Добавляли приблизительно 50 мкл очищенной дистиллированной воды без РНКазы и измеряли количество и чистоту РНК с использованием спектрофотометра. Для синтеза кДНК 2 мкг очищенной тотальной РНК подвергали реакции связывания с олиго-dT при 70°C в течение 5 минут. Затем добавляли 10-кратный буферный раствор для реакции обратной транскрипции, 10 мМ dNTP, ингибитор РНКазы и обратной транскриптазы M-MLV (Enzynomics, Korea), и осуществляли реакцию синтеза кДНК при 42°C в течение 60 минут. Затем, обратную транскриптазу инактивировали посредством нагревания при 72°C в течение 5 минут, а затем добавляли РНКазу H для удаления одноцепочечной РНК, которую использовали в качестве матрицы для полимеразной цепной реакции гена p53.
Для получения из дермальных фибробластов человека гена p53, из которого удаляли последовательность сигнального пептида, синтезировали праймер (T2p53), кодирующий от амино-концевой глутаминовой кислоты, и праймер (Xp53), кодирующий от карбокси-конца, а затем осуществляли ПЦР с использованием кДНК, полученной, как описано выше, в качестве матрицы. Последовательность каждого праймера представлена в таблице 1 ниже.
[Таблица 1]
0,2 пмоль праймера T2p53 и 0,2 пмоль праймера Xp53 смешивали с 0,2 нМ dNTP, 1-кратным реакционным буферным раствором для ДНК-полимеразы AccuPrime Taq (Invitrogen, USA) и 1 ед. ДНК-полимеразы AccuPrime Taq. Затем в устройстве для полимеразной цепной реакции осуществляли реакции амплификации при 95°C в течение 40 секунд, 58°C в течение 30 секунд, 72°C в течение 1 минуты в течение 40 циклов. После реакции амплифицированный фрагмент ДНК приблизительно 1,2 т.п.н. выделяли посредством электрофореза на 1% агарозном геле, а затем встраивали в вектор pGEM-T easy (Promega, USA) с использованием ДНК-лигазы T4. В результате секвенирования полученной таким образом ДНК подтверждали получение кДНК, кодирующей белок p53 человека. Полученный ген p53 обозначали как pTA-p53, и его последовательность оснований является той же, что и последовательность оснований SEQ ID NO: 3 (фигура 1).
Пример 1.2. Получение экспрессирующего вектора E. coli для p53
Пример 1.2.1. Получение плазмиды pET15b-UB-p53
Для получения белка p53 в форме, с которой слит убиквитин, получали следующий экспрессирующий вектор. Для получения гена убиквитина получали праймер NdeUB и праймер T2UB. Последовательность каждого праймера приведена в таблице 2 ниже.
[Таблица 2]
0,2 пмоль праймера NdeUB и 0,2 пмоль праймера T2UB смешивали с 0,2 нМ dNTP, 1-кратным реакционным буферным раствором для ДНК-полимеразы AccuPrime Taq (Invitrogen, USA) и 1 ед. ДНК-полимеразы AccuPrime Taq. Затем в устройстве для полимеразной цепной реакции осуществляли реакции амплификации при 95°C в течение 40 секунд, 58°C в течение 30 секунд, 72°C в течение 1 минуты в течение 25 циклов для получения гена убиквитина (UB). Амплифицированный ген убиквитина расщепляли с помощью ферментов рестрикции NdeI и SacII и плазмиду pTA-p53 расщепляли с помощью ферментов рестрикции SacII и XhoI. Затем фрагменты ДНК размером приблизительно 210 п.н. и 1200 п.н. получали посредством электрофореза в 2% агарозном геле, соответственно, а затем встраивали в вектор pET15b, расщепленный с помощью ферментов рестрикции NdeI и XhoI с использованием ДНК-лигазы T4 для получения плазмиды pET15b-UB-p53 (фигура 2). В этом случае UB-p53 представлен последовательностью оснований SEQ ID NO: 6.
Штамм E. coli BL21 (DE3) трансформировали с использованием плазмиды pET15b-UB-p53. Затем трансформированный штамм культивировали в твердой среде Лурия-Бертани (LB), в которую добавляли антибиотик ампициллин, а затем полученные колонии культивировали в жидкой среде LB при 37°C. Затем, когда плотность клеток достигала оптической плотности приблизительно 0,2 при OD600, добавляли IPTG для достижения конечной концентрации 1 мМ, а затем осуществляли культивирование при встряхивании еще в течение приблизительно 4 часов.
Часть клеток E. coli получали посредством центрифугирования, а затем клетки разрушали и осуществляли электрофорез в ПААГ с SDS. Как показано на фигуре 3, подтверждали экспрессию слитого с убиквитином белка p53, имеющего размер приблизительно 60 кДа. В этом случае дорожка M на фигуре 3 соответствует маркеру молекулярной массы белков, и дорожка 1 соответствует преципитату, центрифугируемому после разрушения E. coli через 4 часа после добавления IPTG, и дорожка 2 соответствует супернатанту, центрифугируемому после разрушения E. coli.
Пример 1.2.2. Получение плазмиды pET11C-TOM70-UB-p53
Для получения белка p53 в форме, с которой слит TOM70, связывающийся с митохондриальной внешней мембраны, и убиквитин, получали экспрессирующий вектор, способный экспрессировать p53 в форме, с которой слиты TOM70 и убиквитин. Для получения генов TOM70 и убиквитина, получали праймер NdeTOM70, праймер TOM70-AS, праймер TOM70UB-S и праймер T2UB-AS. Последовательность каждого праймера приведена в таблице 3 ниже.
[Таблица 3]
Для получения гена TOM70 0,2 пмоль праймера NdeTOM70 и 0,2 пмоль праймера TOM70-AS смешивали с 0,2 нМ dNTP, 1-кратным реакционным буферным раствором для ДНК-полимеразы AccuPrime Taq (Invitrogen, USA) и 1 ед. ДНК-полимеразы AccuPrime Taq. Затем для получения гена TOM70 в устройстве для полимеразной цепной реакции осуществляли реакции амплификации при 95°C в течение 40 секунд, 58°C в течение 30 секунд, 72°C в течение 1 минуты в течение 25 циклов. Амплифицированный фрагмент ДНК обозначали как N-TOM70. Плазмиду pET15b-UB-p53, полученную в примере 1.2.1. выше, использовали в качестве матрицы, добавляли 0,2 пмоль праймера TOM70UB-S и 0,2 пмоль праймера T2UB-AS и смешивали с 0,2 нМ dNTP, 1-кратным реакционным буферным раствором для ДНК-полимеразы AccuPrime Taq (Invitrogen, USA) и 1 ед. ДНК-полимеразы AccuPrime Taq. Затем для получения гена UB в устройстве для полимеразной цепной реакции осуществляли реакции амплификации при 95°C в течение 40 секунд, 58°C в течение 30 секунд, 72°C в течение 1 минуты в течение 25 циклов. Амплифицированный фрагмент ДНК обозначали как C-UB.
Амплифицированную ДНК N-TOM70 и C-UB использовали в качестве матрицы, и 0,2 пмоль праймера NdeTOM70, 0,2 пмоль праймера T2UB-AS смешивали с 0,2 нМ dNTP, 1-кратным реакционным буферным раствором для ДНК-полимеразы AccuPrime Taq (Invitrogen, USA) и 1 ед. ДНК-полимеразы AccuPrime Taq. Затем для получения гена убиквитина TOM70-UB, с которым слит амплифицированный TOM70, в устройстве для полимеразной цепной реакции осуществляли реакции амплификации при 95°C в течение 40 секунд, 58°C в течение 30 секунд, 72°C в течение 1 минуты в течение 25 циклов.
Амплифицированный ген TOM70-UB расщепляли с помощью ферментов рестрикции NdeI и SacII, плазмиду pTA-p53 расщепляли с помощью SacII и XhoI и получали фрагменты ДНК размером 330 п.н. и 1500 п.н. посредством электрофореза в 2% агарозном геле, соответственно. Затем, их встраивали в вектор pET11c, расщепленный с помощью ферментов рестрикции NdeI и SalI, с использованием ДНК-лигазы T4 для получения плазмиды pET11c-TOM70-UB-p53 (фигура 4). В этом случае TOM70-UB-p53 представлен последовательностью оснований SEQ ID NO: 11.
Штамм E. coli BL21 (DE3) трансформировали с использованием плазмиды pET11c-TOM70-UB-p53. Затем, трансформированный штамм культивировали в твердой среде Лурия-Бертани (LB), в которую добавляли антибиотик ампициллин, а затем полученные колонии культивировали в жидкой среде LB в инкубаторе-встряхивателе при 37°C. Затем, когда плотность клеток достигала оптической плотности приблизительно 0,2 при OD600, добавляли IPTG для достижения конечной концентрации 1 мМ, а затем осуществляли культивирование при встряхивании еще в течение приблизительно 4 часов.
Часть клеток E. coli получали посредством центрифугирования, а затем клетки разрушали и осуществляли электрофорез в ПААГ с SDS. Как показано на фигуре 5, подтверждали, что экспрессировали белок p53, имеющий размер приблизительно 62 кДа, в форме, с которой слиты TOM70 и убиквитин. В этом случае дорожка M соответствует маркеру молекулярной массы белков, и дорожка 1 соответствует супернатанту, центрифугируемому после разрушения E. coli через 4 часа после добавления IPTG.
Пример 1.2.3. Получение плазмиды pET11c-TOM70-(GGGGS)3-UB-p53
Для получения белка p53 в форме, с которой слиты TOM70, связывающийся с митохондриальной внешней мембраной, линкер (GGGGSGGGGSGGGGS (SEQ ID NO: 70)) и убиквитин, получали экспрессирующий вектор, способный экспрессировать белок p53 в форме, с которой слиты TOM70, линкер и убиквитин. Для получения гена линкера, связанного с TOM70, получали праймер TOM70(G)3-AS, праймер (G)3UB-S и праймер Xp53 (noT). Последовательность каждого праймера приведена в таблице 4 ниже.
[Таблица 4]
Плазмиду pET11c-TOM70-UB-p53, полученную в примере 1.2.2. выше, использовали в качестве матрицы, и добавляли 0,2 пмоль праймера NdeTOM70 и 0,2 пмоль праймера TOM70(G)3-AS, и смешивали с 0,2 нМ dNTP, 1-кратным реакционным буферным раствором для ДНК-полимеразы AccuPrime Taq (Invitrogen, USA) и 1 ед. ДНК-полимеразы AccuPrime Taq. Затем для получения гена TOM70-G3, в котором связаны ген TOM70 и линкер, в устройстве для полимеразной цепной реакции осуществляли реакции амплификации при 95°C в течение 40 секунд, 58°C в течение 30 секунд, 72°C в течение 1 минуты в течение 25 циклов. Кроме того, плазмиду pET15b-UB-p53, полученную в примере 1.2.1. выше, использовали в качестве матрицы и смешивали 0,2 пмоль праймера (G)3UB-S и 0,2 пмоль праймера Xp53 (noT) с 0,2 нМ dNTP, 1-кратным реакционным буферным раствором для ДНК-полимеразы AccuPrime Taq (Invitrogen, USA) и 1 ед. ДНК-полимеразы AccuPrime Taq.
Затем для получения UB-p53, p53, слитого с геном убиквитина, в устройстве для полимеразной цепной реакции осуществляли реакции амплификации при 95°C в течение 40 секунд, 58°C в течение 30 секунд, 72°C в течение 1 минуты в течение 25 циклов. Амплифицированный ген TOM70-G3 расщепляли с помощью ферментов рестрикции NdeI и BamHI, амплифицированный ген UB-p53 расщепляли с помощью BamHI и XhoI и фрагменты ДНК размером 100 п.н. и 1500 п.н. получали посредством электрофореза в 2% агарозном геле, соответственно. Затем их встраивали в вектор pET11c, расщепленный с помощью ферментов рестрикции NdeI и SalI с использованием ДНК-лигазы T4, для получения плазмиды pET11c-TOM70-(GGGGS)3-UB-p53 (фигура 6). В этом случае TOM70-(GGGGS)3-UB-p53 представлен последовательностью оснований SEQ ID NO: 15.
Штамм E. coli BL21 (DE3) трансформировали с использованием плазмиды pET11c-TOM70-(GGGGS)3-UB-p53. Затем трансформированный штамм культивировали в твердой среде Лурия-Бертани (LB), в которую добавляли антибиотик ампициллин, а затем полученные колонии культивировали в жидкой среде LB при 37°C. Затем, когда плотность клеток достигала оптической плотности приблизительно 0,2 при OD600, добавляли IPTG для достижения конечной концентрации 1 мМ, а затем осуществляли культивирование при встряхивании еще в течение приблизительно 4 часов.
Часть клеток E. coli получали посредством центрифугирования, а затем клетки разрушали и осуществляли электрофорез в ПААГ с SDS. Как показано на фигуре 7, подтверждали, что экспрессировали белок p53, имеющий размер приблизительно 62 кДа, в форме, с которой слиты TOM70, линкер и убиквитин. В этом случае дорожка M соответствует маркеру молекулярной массы белков, дорожка 1 соответствует преципитату, центрифугируемому после разрушения E. coli через 4 часа после добавления IPTG, и дорожка 2 соответствует супернатанту, центрифугируемому после разрушения E. coli.
Пример 1.2.4. Получение плазмиды pET11c-TOM70-(GGGGS)3-p53
Для получения белка p53 в форме, с которой слиты TOM70, связывающийся с митохондриальной внешней мембраной, и линкер (GGGGSGGGGSGGGGS), получали экспрессирующий вектор, способный экспрессировать белок p53 в форме, с которой слиты TOM70 и линкер. Для получения гена p53, с которым слиты TOM70 и линкер, получали праймер (B(G)3p53). Последовательность каждого праймера приведена в таблице 5 ниже.
[Таблица 5]
Плазмиду pET11c-TOM70-UB-p53, полученную в примере 1.2.2. выше, использовали в качестве матрицы, добавляли 0,2 пмоль праймера NdeTOM70 и 0,2 пмоль праймера TOM70(G)3-AS и смешивали с 0,2 нМ dNTP, 1-кратным реакционным буферным раствором для ДНК-полимеразы AccuPrime Taq (Invitrogen, USA) и 1 ед. ДНК-полимеразы AccuPrime Taq. Затем для получения гена TOM70 в устройстве для полимеразной цепной реакции осуществляли реакции амплификации при 95°C в течение 40 секунд, 58°C в течение 30 секунд, 72°C в течение 1 минуты в течение 25 циклов. Амплифицированный фрагмент ДНК обозначали как TOM70-G3.
Плазмиду pET15b-UB-p53, полученную в примере 1.2.1. выше, использовали в качестве матрицы, и смешивали 0,2 пмоль праймера B(G)3p53 и 0,2 пмоль праймера Xp53 (noT) с 0,2 нМ dNTP, 1-кратным реакционным буферным раствором для ДНК-полимеразы AccuPrime Taq (Invitrogen, USA) и 1 ед. ДНК-полимеразы AccuPrime Taq. Затем в устройстве для полимеразной цепной реакции осуществляли реакции амплификации при 95°C в течение 40 секунд, 58°C в течение 30 секунд, 72°C в течение 1 минуты в течение 25 циклов. Амплифицированный фрагмент ДНК обозначали как G3-p53.
Амплифицированный фрагмент ДНК, TOM70-G3, расщепляли с помощью NdeI и BamHI и фрагмент ДНК G3-53 расщепляли с помощью ферментов рестрикции BamHI и XhoI. Затем фрагменты ДНК размером приблизительно 150 п.н. и 1300 п.н. получали посредством электрофореза в 2% агарозном геле, соответственно, а затем встраивали в вектор pET11c, расщепленный с помощью ферментов рестрикции NdeI и SalI с использованием ДНК-лигазы T4 для получения плазмиды pET11c-TOM70-(GGGGS)3-p53 (фигура 8). В этом случае TOM70-(GGGGS)3-p53 представлен последовательностью оснований SEQ ID NO: 17.
Штамм E. coli BL21(DE3) трансформировали с использованием плазмиды pET11c-TOM70-(GGGGS)3-p53. Затем трансформированный штамм культивировали в твердой среде Лурия-Бертани (LB), в которую добавляли антибиотик ампициллин, а затем полученные колонии культивировали в жидкой среде LB в инкубаторе-встряхивателе при 37°C. Затем, когда плотность клеток достигала оптической плотности приблизительно 0,2 при OD600, добавляли IPTG для достижения конечной концентрации 1 мМ, а затем осуществляли культивирование при встряхивании еще в течение приблизительно 4 часов.
Часть клеток E. coli получали посредством центрифугирования, а затем клетки разрушали и осуществляли электрофорез в ПААГ с SDS. Как показано на фигуре 9, подтверждали экспрессию белка p53, имеющего размер приблизительно 60 кДа, в форме, с которой слит TOM70. В этом случае дорожка M соответствует маркеру молекулярной массы белков, дорожка 1 соответствует преципитату, центрифугируемому после разрушения E. coli через 4 часа после добавления IPTG, и дорожка 2 соответствует супернатанту, центрифугируемому после разрушения E. coli.
Пример 1.2.5. pET15b-UB-p53-TOM7
Для получения белка p53 в форме, с которой слиты убиквитин и TOM7, связывающийся с митохондриальной внешней мембраной, получали экспрессирующий вектор, способный экспрессировать p53 в форме, с которой слиты убиквитин, p53 и TOM. Для получения гена p53, с которым слиты TOM7 и убиквитин, получали праймер Xp53(noT), праймер XTOM7 и праймер LTOM7. Последовательность каждого праймера приведена в таблице 6 ниже.
[Таблица 6]
Плазмиду pET15b-UB-p53, полученную в примере 1.2.1. выше, использовали в качестве матрицы, добавляли 0,2 пмоль праймера NdeUB и 0,2 пмоль праймера Xp53(noT) и смешивали с 0,2 нМ dNTP, 1-кратным реакционным буферным раствором для ДНК-полимеразы AccuPrime Taq (Invitrogen, USA) и 1 ед. ДНК-полимеразы AccuPrime Taq. Затем для получения гена UB-p53 в устройстве для полимеразной цепной реакции осуществляли реакции амплификации при 95°C в течение 40 секунд, 58°C в течение 30 секунд, 72°C в течение 1 минуты в течение 25 циклов. Кроме того, полученную, как описано выше, кДНК использовали в качестве матрицы, и смешивали 0,2 пмоль праймера XTOM7 и 0,2 пмоль праймера LTOM7 с 0,2 нМ dNTP, 1-кратным реакционным буферным раствором для ДНК-полимеразы AccuPrime Taq (Invitrogen, USA) и 1 ед. ДНК-полимеразы AccuPrime Taq.
Затем для получения гена TOM7 в устройстве для полимеразной цепной реакции осуществляли реакции амплификации при 95°C в течение 40 секунд, 58°C в течение 30 секунд, 72°C в течение 1 минуты в течение 40 циклов. Амплифицированный фрагмент ДНК, UB-p53, расщепляли с помощью ферментов рестрикции NdeI и XhoI и амплифицированный ген TOM7 расщепляли с помощью ферментов рестрикции XhoI и SalI. Фрагменты ДНК размером приблизительно 1500 п.н. и 150 п.н. получали посредством электрофореза в 2% агарозном геле, соответственно, а затем встраивали в вектор pET15b, расщепленный с помощью ферментов рестрикции NdeI и XhoI, с использованием ДНК-лигазы T4 для получения плазмиды pET15b-UB-p53-TOM7 (фигура 10). В этом случае UB-p53-TOM7 представлен последовательностью оснований SEQ ID NO: 21.
Штамм E. coli BL21 (DE3) трансформировали с использованием плазмиды pET15b-UB-p53-TOM7. Затем трансформированный штамм культивировали в твердой среде Лурия-Бертани (LB), в которую добавляли антибиотик ампициллин, а затем полученные колонии культивировали в жидкой среде LB при 37°C. Затем, когда плотность клеток достигала оптической плотности приблизительно 0,2 при OD600, добавляли IPTG для достижения конечной концентрации 1 мМ, а затем осуществляли культивирование при встряхивании еще в течение приблизительно 4 часов.
Часть клеток E. coli получали посредством центрифугирования, а затем клетки разрушали и осуществляли электрофорез в ПААГ с SDS. Как показано на фигуре 11, подтверждали экспрессию белка p53, имеющего размер приблизительно 60 кДа, в форме, с которой слиты убиквитин и TOM7. В этом случае дорожка M соответствует маркеру молекулярной массы белков, дорожка 1 соответствует преципитату, центрифугируемому после разрушения E. coli через 4 часа после добавления IPTG, и дорожка 2 соответствует супернатанту, центрифугируемому после разрушения E. coli.
Пример 1.2.6. Конструирование экспрессирующего вектора млекопитающих pCMV-p53-myc/His
Получали экспрессирующий вектор для клеток животных, способный экспрессировать p53. Для получения гена p53 получали праймер Rp53. Последовательность каждого праймера приведена в таблице 7 ниже.
[Таблица 7]
Плазмиду pET-UB-p53, полученную в примере 1.2.1. выше, использовали в качестве матрицы и смешивали 0,2 пмоль праймера Rp53 и 0,2 пмоль праймера Xp53(noT) с 0,2 нМ dNTP, 1-кратным реакционным буферным раствором для ДНК-полимеразы AccuPrime Taq (Invitrogen, USA) и 1 ед. ДНК-полимеразы AccuPrime Taq. Затем для получения гена p53 в устройстве для полимеразной цепной реакции осуществляли реакции амплификации при 95°C в течение 40 секунд, 58°C в течение 30 секунд, 72°C в течение 1 минуты в течение 25 циклов.
Амплифицированный ген p53 расщепляли с помощью ферментов рестрикции EcoRI и XhoI, и фрагмент ДНК размером приблизительно 1300 п.н. получали посредством электрофореза в 2% агарозном геле, а затем его встраивали в вектор pcDNA3.1-myc/His A, расщепленный с помощью ферментов рестрикции EcoRI и XhoI, с использованием ДНК-лигазы T4 для получения плазмиды pCMV-p53-myc/His (фигура 12). В этом случае p53-myc/His представлен последовательностью оснований SEQ ID NO: 23.
Клетки животных CHO трансфицировали с использованием плазмиды pCMV-p53-myc/His, клетки разрушали, а затем осуществляли электрофорез в ПААГ с SDS и анализировали посредством вестерн-блоттинга с использованием антитела против c-myc. Как показано на фигуре 13, подтверждали экспрессию белка p53, имеющего размер приблизительно 55 кДа. В этом случае дорожка M соответствует маркеру молекулярной массы белков, и на дорожке 1 видно, что осуществляли трансфекцию клеток животных CHO, и клетки разрушали, а затем осуществляли электрофорез в ПААГ с SDS, а затем подтверждали посредством вестерн-блоттинга с использованием антитела против c-myc.
Пример 1.3. Выделение и очистка слитого белка, содержащего p53
Пример 1.3.1. Выделение и очистка рекомбинантного белка TOM70-(GGGGS)3-p53, полученного из E. coli
Производственный штамм E. coli BL21(DE3), экспрессирующий рекомбинантный белок TOM70-(GGGGS)3-p53, инокулировали в жидкую среду LB и культивировали при 37°C. Затем, когда оптическая плотность достигала 0,4 при OD600, добавляли 0,5 мМ IPTG, и осуществляли культивирование при встряхивании еще в течение 4 часов для экспрессии белка TOM70-(GGGGS)3-p53.
После завершения культивирования клетки выделяли посредством центрифугирования и выделенные клетки промывали однократно с использованием PBS, а затем клетки суспендировали с использованием раствора PBS и разрушали суспендированные клетки с использованием ультразвукового гомогенизатора. Разрушенные клетки центрифугировали с использованием высокоскоростной центрифуги, а затем выделяли нерастворимые фракции и промывали выделенные нерастворимые фракции три раза с использованием 50 мМ Трис, 100 мМ раствора этилендиаминтетрауксусной кислоты (ЭДТА), pH 8,0. Затем их растворяли в растворе 6 M гуанидина, 100 мМ фосфата натрия, 10 мМ Трис, pH 8,0, и фильтровали с использованием фильтра 0,45 мкм, а затем нагружали на упакованную никелевую колонку для хроматографии для осуществления первичной очистки.
Нагружали раствор, содержащий белок TOM70-(GGGGS)3-p53, а затем, пропускали промывочный раствор 8 М мочевины, 50 мМ фосфата натрия, 500 мМ NaCl, 10 мМ имидазола, pH 8,0, до тех пор, пока несвязанные примеси не будут неопределимыми, и элюировали белок с использованием 8 М мочевины, 50 мМ фосфата натрия, 500 мМ NaCl, 500 мМ имидазола, pH 8,0, изменяя концентрацию имидазола до 50 мМ, 100 мМ, 250 мМ, 500 мМ (фигура 14). В этом случае дорожка M на фигуре 14 соответствует маркеру молекулярной массы белков, и дорожка 1 соответствует нагруженному образцу для никель-аффинной хроматографии. На дорожке 2 видно, что он не связывался с никель-аффинной смолой. Дорожки 3-4 соответствуют результатам элюции с использованием раствора 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/50 мМ имидазола. Дорожки 5-7 соответствуют результатам элюции с использованием раствора 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/100 мМ имидазола. Дорожки 8-9 соответствуют результатам элюции с использованием раствора 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/250 мМ имидазола. Дорожки 10-11 соответствуют результатам элюции с использованием раствора 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/500 мМ имидазола.
Элюированный раствор, выделенный при хроматографии на никеле, подвергали замене буфера с помощью PBS с использованием принципа осмотического давления. После завершения замены буфера элюированный раствор центрифугировали для выделения супернатанта, и количество белка в выделенном элюированном растворе измеряли способом количественного анализа белков и подтверждали с использованием электрофореза в ПААГ с SDS. Как показано на фигуре 15, после подтверждения белок TOM70-(GGGGS)3-p53 быстро охлаждали жидким азотом и хранили в криогенном морозильном устройстве при -80°C. В этом случае дорожка M соответствует маркеру молекулярной массы белков, и дорожка 1 соответствует белку TOM70-(GGGGS)3-p53, полученному после диализа в буферном растворе PBS.
Пример 1.3.2. Выделение и очистка рекомбинантного белка TOM70-(GGGGS)3-UB-p53, полученного из E. coli
E. coli, экспрессирующие рекомбинантный белок TOM70-(GGGGS)3-UB-p53, использовали для выделения и очистки белка TOM70-(GGGGS)3-UB-p53 тем же способом, что и в примере 1.3.1. В результате элюировали белок TOM70-(GGGGS)3-UB-p53 (фигура 16). В этом случае дорожка M на фигуре 16 соответствует маркеру молекулярной массы белков, и дорожка 1 соответствует нагруженному образцу для никель-аффинной хроматографии. На дорожке 2 видно, что он не связан с никель-аффинной смолой. Дорожка 3 соответствует результатам элюции с использованием раствора 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/50 мМ имидазола. Дорожки 4-7 соответствуют результатам элюции с использованием раствора 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/100 мМ имидазола. Дорожки 8-11 соответствуют результатам элюции с использованием раствора 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/250 мМ имидазола.
Количество белка в выделенном элюированном растворе измеряли способом количественного анализа белков и подтверждали с использованием электрофореза в ПААГ с SDS. Как показано на фигуре 17, после подтверждения белок TOM70-(GGGGS)3-UB-p53 быстро охлаждали жидким азотом и хранили в криогенном морозильном устройстве при -80°C. В этом случае дорожка M на фигуре 17 соответствует маркеру молекулярной массы белков, и на дорожке 1 показан белок TOM70-(GGGGS)3-UB-p53, полученный после диализа в буферном растворе PBS.
Пример 1.3.3. Выделение и очистка рекомбинантного белка UB-p53, полученного из E. coli
Производственный штамм BL21 (DE3), экспрессирующий белок UB-p53 в зрелой форме, с которой слит убиквитин, инокулировали в жидкую среду LB и культивировали в инкубаторе-встряхивателе при 37°C. Когда оптическая плотность достигала 0,4 при OD600, добавляли 0,5 мМ IPTG и осуществляли культивирование при встряхивании еще в течение 4 часов для экспрессии белка UB-p53 в зрелой форме, с которой слит убиквитин.
Затем белок UB-p53 выделяли и очищали тем же способом, что и в примере 1.3.1. В результате элюировали белок UB-p53 (фигура 18). В этом случае дорожка M на фигуре 18 соответствует маркеру молекулярной массы белков, и дорожка 1 соответствует нагруженному образцу для никель-аффинной хроматографии. На дорожке 2 видно, что он не связан с никель-аффинной смолой. Дорожка 3 соответствует результатам элюции с использованием раствора 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/50 мМ имидазола. Дорожки 4-6 соответствуют результатам элюции с использованием раствора 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/100 мМ имидазола. Дорожки 7-9 соответствуют результатам элюции с использованием раствора 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/250 мМ имидазола. Дорожки 10-11 соответствуют результатам элюции с использованием раствора 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/500 мМ имидазола.
Количество белка в выделенном элюированном растворе измеряли способом количественного анализа белков и подтверждали с использованием электрофореза в ПААГ с SDS. Как показано на фигуре 19, после подтверждения белок UB-p53 быстро охлаждали жидким азотом и хранили в криогенном морозильном устройстве при -80°C. В этом случае дорожка M на фигуре 19 соответствует маркеру молекулярной массы белков, и на дорожке 1 показан белок UB-p53, полученный после диализа в буферном растворе PBS.
Пример 1.3.4. Выделение и очистка рекомбинантного белка UB-p53-TOM7, полученного из E. coli
Производственный штамм E. coli BL21 (DE3), экспрессирующий белок UB-p53-TOM7 в зрелой форме, с которой слит убиквитин, инокулировали в жидкую среду LB и культивировали при 37°C. Когда оптическая плотность достигала 0,4 при OD600, добавляли 0,5 мМ IPTG и осуществляли культивирование при встряхивании еще в течение 4 часов для экспрессии белка UB-p53-TOM7 в зрелой форме, с которой слит убиквитин.
Затем белок UB-p53-TOM7 выделяли и очищали тем же способом, что и в примере 1.3.1. В результате элюировали белок UB-p53-TOM7 (фигура 20). В этом случае дорожка M на фигуре 20 соответствует маркеру молекулярной массы белков, и дорожка 1 соответствует нагруженному образцу для никель-аффинной хроматографии. На дорожке 2 видно, что он не связан с никель-аффинной смолой. Дорожка 3 соответствует результатам элюции с использованием раствора 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/10 мМ имидазола. Дорожка 4 соответствует результатам элюции с использованием раствора 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/50 мМ имидазола. Дорожки 5-7 соответствуют результатам элюции с использованием раствора 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/100 мМ имидазола. Дорожки 8-9 соответствует результатам элюции с использованием раствора 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/250 мМ имидазола. Дорожки 10-11 соответствуют результатам элюции с использованием раствора 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/500 мМ имидазола.
Количество белка в выделенном элюированном растворе измеряли способом количественного анализа белков и подтверждали с использованием электрофореза в ПААГ с SDS. Как показано на фигуре 21, после подтверждения белок UB-p53 быстро охлаждали жидким азотом и хранили в криогенном морозильном устройстве при -80°C. В этом случае дорожка M на фигуре 21 соответствует маркеру молекулярной массы белков, и на дорожке 1 показан белок UB-p53-TOM7, полученный после диализа в буферном растворе PBS.
Пример 2. Получение слитого белка, содержащего гранзим B
Пример 2.1. Амплификация гена гранзима B
Для экспрессии гранзима B человека в рекомбинантном белке выделяли тотальную РНК из естественных киллеров человека и синтезировали из нее кДНК. В частности, естественные киллеры человека культивировали в среде с 10% сыворотки в условиях 5% диоксида углерода и 37°C (1×106 клеток). Затем получали РНК тем же способом, что и в примере 1.1., а затем ее использовали в качестве матрицы для полимеразной цепной реакции гена гранзима B.
Для получения гена гранзима B, из которого в естественных киллерах человека удалена последовательность сигнального пептида, синтезировали праймер T2GZMB, кодирующий от амино-концевого изолейцина, и праймер XGZMB(noT), кодирующий с карбокси-конца, а затем осуществляли ПЦР с использованием кДНК, полученной, как описано выше, в качестве матрицы. Последовательность каждого праймера приведена в таблице 8 ниже.
[Таблица 8]
кДНК, полученную, как описано выше, использовали в качестве матрицы и смешивали 0,2 пмоль праймера T2GZMB и 0,2 пмоль праймера XGZMB(noT) с 0,2 нМ dNTP, 1-кратным реакционным буферным раствором для ДНК-полимеразы AccuPrime Taq (Invitrogen, USA) и 1 ед. ДНК-полимеразы AccuPrime Taq. Затем в устройстве для полимеразной цепной реакции осуществляли реакции амплификации при 95°C в течение 40 секунд, 58°C в течение 30 секунд, 72°C в течение 1 минуты в течение 40 циклов. После реакции амплифицированный фрагмент ДНК размером приблизительно 700 п.н. выделяли посредством электрофореза в 1% агарозном геле, а затем встраивали в вектор pGEM-T easy (Promega, USA) с использованием ДНК-лигазы T4. В результате секвенирования полученной таким образом ДНК подтверждали получение кДНК, кодирующей белок гранзим B человека. Полученный ген гранзима B обозначали как pTA-гранзим B, и ген гранзима B представлен последовательностью оснований SEQ ID NO: 26 (фигура 22).
Пример 2.2. Получение экспрессирующего вектора E. coli для белка гранзима B
Пример 2.2.1. Получение плазмиды pET11c-TOM70-(GGGGS)3-UB-гранзим B
Для получения белка гранзима B в форме, с которой слиты TOM70, связывающийся с митохондриальной внешней мембраной, линкер (GGGGSGGGGSGGGGS) и убиквитин, получали экспрессирующий вектор, способный экспрессировать гранзим B в форме, с которой слиты TOM70, линкер и убиквитин.
Плазмиду pTA-гранзим B, полученную в примере 2.1. выше, расщепляли с помощью ферментов рестрикции SacII и XhoI, и фрагмент ДНК размером приблизительно 700 п.н. получали посредством электрофореза в 2% агарозном геле. Затем его встраивали в вектор pET11c-TOM70-(GGGGS)3-UB-(p53), расщепленный с помощью ферментов рестрикции SacII и XhoI, с использованием ДНК-лигазы T4 для получения плазмиды pET11c-TOM70-(GGGGS)3-UB-гранзим B (SEQ ID NO: 27) (фигура 23).
Штамм E. coli BL21 (DE3) трансформировали с использованием плазмиды pET11c-TOM70-(GGGGS)3-UB-гранзим B. Затем трансформированный штамм культивировали в твердой среде Лурия-Бертани (LB), в которую добавляли антибиотик ампициллин, а затем полученные колонии культивировали в жидкой среде LB в инкубаторе-встряхивателе при 37°C. Затем, когда плотность клеток достигала оптической плотности приблизительно 0,2 при OD600, добавляли IPTG для достижения конечной концентрации 0,5 мМ, а затем осуществляли культивирование при встряхивании еще в течение приблизительно 4 часов.
Часть клеток E. coli получали посредством центрифугирования, а затем клетки разрушали и осуществляли электрофорез в ПААГ с SDS. Как показано на фигуре 24, подтверждали, что экспрессировали белок гранзим B, имеющий размер приблизительно 35 кДа, в форме, с которой слиты TOM70, линкер и убиквитин. В этом случае дорожка M на фигуре 24 соответствует маркеру молекулярной массы белков, дорожка 1 соответствует преципитату, центрифугируемому после разрушения E. coli через 4 часа после добавления IPTG, и дорожка 2 соответствует супернатанту, центрифугируемому после разрушения E. coli.
Пример 2.2.2. Получение плазмиды pET15b-UB-гранзим B-TOM7
Для получения белка гранзима B в форме, с которой слиты убиквитин и TOM7, связывающийся с митохондриальной внешней мембраной, получали экспрессирующий вектор, способный экспрессировать белок гранзим B в форме, с которой слиты убиквитин, гранзим B и TOM7.
Плазмиду pTA-гранзим B, полученную в примере 2.1. выше, расщепляли с помощью ферментов рестрикции SacII и XhoI, и фрагмент ДНК размером приблизительно 700 п.н. получали посредством электрофореза в 2% агарозном геле. Затем его встраивали в вектор pET15b-UB-(p53)-TOM7, расщепленный с помощью ферментов рестрикции SacII и XhoI, с использованием ДНК-лигазы T4 для получения плазмиды pET15b-UB-гранзим B-TOM7 (фигура 25). В этом случае, UB-гранзим B-TOM7 представлен последовательностью оснований SEQ ID NO: 28.
Штамм E. coli BL21 (DE3) трансформировали с использованием плазмиды pET15b-UB-гранзим B-TOM7. Затем трансформированный штамм культивировали в твердой среде Лурия-Бертани (LB) в которую добавляли антибиотик ампициллин, а затем полученные колонии культивировали в жидкой среде LB при 37°C. Затем, когда плотность клеток достигала оптической плотности приблизительно 0,2 при OD600, добавляли IPTG для достижения конечной концентрации 0,5 мМ, а затем осуществляли культивирование при встряхивании еще в течение приблизительно 4 часов.
Часть клеток E. coli получали посредством центрифугирования, а затем клетки разрушали и осуществляли электрофорез в ПААГ с SDS. Как показано на фигуре 26, подтверждали, что экспрессировали белок гранзим B, имеющий размер приблизительно 35 кДа, в форме, с которой слиты убиквитин и TOM70. В этом случае дорожка M на фигуре 26 соответствует маркеру молекулярной массы белков, дорожка 1 соответствует преципитату, центрифугируемому после разрушения E. coli через 4 часа после добавления IPTG, и дорожка 2 соответствует супернатанту, центрифугируемому после разрушения E. coli.
Пример 2.3. Выделение и очистка рекомбинантного белка TOM70-(GGGGS)3-UB-гранзим B, полученного из E. coli
Белок TOM70-(GGGGS)3-UB-гранзим B выделяли и очищали тем же способом, что и в примере 1.3.1. В результате элюировали белок TOM70-(GGGGS)3-UB-гранзим B (фигура 27). В этом случае дорожка M на фигуре 27 соответствует маркеру молекулярной массы белков, и дорожка 1 соответствует нагруженному образцу для никель-аффинной хроматографии. На дорожке 2 видно, что он не связан с никель-аффинной смолой. Дорожки 3 и 4 соответствуют результатам элюции с использованием раствора 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/50 мМ имидазола. Дорожки 5-7 соответствуют результатам элюции с использованием раствора 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/100 мМ имидазола. Дорожки 8-9 соответствуют результатам элюции с использованием раствора 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/250 мМ имидазола.
Количество белка в выделенном элюированном растворе измеряли способом количественного анализа белков и подтверждали с использованием электрофореза в ПААГ с SDS. Как показано на фигуре 28, после подтверждения белок TOM70-(GGGGS)3-UB-гранзим B быстро охлаждали жидким азотом и хранили в криогенном морозильном устройстве при -80°C. В этом случае дорожка M на фигуре 28 соответствует маркеру молекулярной массы белков, и на дорожке 1 показан белок TOM70-(GGGGS)3-UB-гранзим B, полученный после диализа в буферном растворе PBS.
Пример 3. Получение слитого белка, содержащего RKIP
Пример 3.1. Амплификация гена RKIP
Для экспрессии гена рекомбинантного белка RKIP человека (ингибиторного белка Raf-киназы) выделяли тотальную РНК из эпителиальных клеток человека и из нее синтезировали кДНК. Дермальные фибробласты человека культивировали в среде с 10% сыворотки в условиях 5% диоксида углерода и 37°C (1×106 клетки). Затем РНК получали тем же способом, что и в примере 1.1., а затем использовали ее в качестве матрицы для полимеразной цепной реакции гена RKIP.
Для получения из дермальных фибробластов человека гена RKIP, из которого удаляли последовательность сигнального пептида, синтезировали праймер T2RKIP, кодирующий с амино-концевого пролина, и праймер XRKIP(noT), кодирующий с карбокси-конца, а затем осуществляли ПЦР с использованием кДНК, полученной, как указано выше, в качестве матрицы. Последовательность каждого праймера приведена в таблице 9 ниже.
[Таблица 9]
кДНК, полученную, как описано выше, использовали в качестве матрицы, и смешивали 0,2 пмоль праймера T2RKIP и 0,2 пмоль праймера XRKIP(noT) с 0,2 нМ dNTP, 1-кратным реакционным буферным раствором для ДНК-полимеразы AccuPrime Taq (Invitrogen, USA) и 1 ед. ДНК-полимеразы AccuPrime Taq. Затем в устройстве для полимеразной цепной реакции осуществляли реакции амплификации при 95°C в течение 40 секунд, 58°C в течение 30 секунд, 72°C в течение 1 минуты в течение 40 циклов. После реакции амплифицированный фрагмент ДНК размером приблизительно 560 п.н. выделяли посредством электрофореза в 1% агарозном геле, а затем встраивали в вектор pGEM-T easy (Promega, USA) с использованием ДНК-лигазы T4. В результате секвенирования полученной таким образом ДНК подтверждали получение кДНК, кодирующей белок RKIP человека. Полученный ген RKIP обозначали как pTA-RKIP (фигура 29), и последовательность оснований гена RKIP представлена последовательностью оснований SEQ ID NO: 31.
Пример 3.2. Получение экспрессирующего вектора E. coli для белка RKIP
Пример 3.2.1. Получение плазмиды pET11c-TOM70-(GGGGS)3-UB-RKIP
Для получения белка RKIP в форме, с которой слиты TOM70, связывающийся с митохондриальной внешней мембраной, линкер (GGGGSGGGGSGGGGS) и убиквитин, получали экспрессирующий вектор, способный экспрессировать RKIP в форме, с которой слиты TOM70, линкер и убиквитин.
Плазмиду pTA-RKIP, полученную в примере 3.1., расщепляли с помощью ферментов рестрикции SacII и XhoI и фрагмент ДНК размером приблизительно 560 п.н. получали посредством электрофореза в 2% агарозном геле, а затем его встраивали в вектор pET11c-TOM70-(GGGGS)3-UB-(p53), расщепленный с помощью ферментов рестрикции SacII и XhoI, с использованием ДНК-лигазы T4 для получения плазмиды pET11-TOM70-(GGGGS)3-UB-RKIP (фигура 30). В этом случае TOM70-(GGGGS)3-UB-RKIP представлен последовательностью оснований SEQ ID NO: 32.
Штамм E. coli BL21(DE3) трансформировали с использованием плазмиды pET11c-TOM70-(GGGGS)3-UB-RKIP. Затем трансформированный штамм культивировали в твердой среде Лурия-Бертани (LB), в которую добавляли антибиотик ампициллин, а затем полученные колонии культивировали в жидкой среде LB в инкубаторе-встряхивателе при 37°C. Затем, когда плотность клеток достигала оптической плотности приблизительно 0,2 при OD600, добавляли IPTG для достижения конечной концентрации 0,5 мМ, а затем осуществляли культивирование при встряхивании еще в течение приблизительно 4 часов.
Часть клеток E. coli получали посредством центрифугирования, а затем клетки разрушали и осуществляли электрофорез в ПААГ с SDS. Как показано на фигуре 31, подтверждали, что экспрессировали белок RKIP, имеющий размер приблизительно 33 кДа, в форме, с которой слиты TOM70, линкер и убиквитин. В этом случае дорожка M на фигуре 31 соответствует маркеру молекулярной массы белков, дорожка 1 соответствует преципитату, центрифугируемому после разрушения E. coli через 4 часа после добавления IPTG, и дорожка 2 соответствует супернатанту, центрифугируемому после разрушения E. coli.
Пример 3.3. Выделение и очистка рекомбинантного белка TOM70-(GGGGS)3-UB-RKIP, полученного из E. coli
Производственный штамм E. coli BL21 (DE3), экспрессирующий рекомбинантный TOM70-(GGGGS)3-UB-RKIP, инокулировали в жидкую среду LB и культивировали при 37°C. Когда оптическая плотность достигала 0,3 при OD600, культуру помещали в холодильную камеру для снижения температуры культурального раствора, и температуру инкубатора изменяли на 18°C, а затем добавляли 0,5 мМ IPTG и осуществляли культивирование при встряхивании еще в течение 1 дня для экспрессии белка TOM70-(GGGGS)3-UB-RKIP.
Затем белок TOM70-(GGGGS)3-UB-RKIP выделяли и очищали тем же способом, что и в примере 1.3.1. В результате элюировали белок TOM70-(GGGGS)3-UB-RKIP (фигура 32). В этом случае дорожка M на фигуре 32 соответствует маркеру молекулярной массы белков, и дорожка 1 соответствует нагруженному образцу для никель-аффинной хроматографии. На дорожке 2 видно, что он не связан с никель-аффинной смолой. Дорожка 3 соответствует результатам элюции с использованием 50 мМ фосфата Na/500 мМ NaCl/10 мМ имидазола. Дорожки 4-6 соответствуют результатам элюции с использованием 50 мМ фосфата Na/500 мМ NaCl/50 мМ имидазола. Дорожки 7-8 соответствуют результатам элюции с использованием 50 мМ фосфата Na/500 мМ NaCl/100 мМ имидазола. Дорожки 9-10 соответствуют результатам элюции с использованием 50 мМ фосфата Na/500 мМ NaCl/175 мМ имидазола. Дорожки 11-13 соответствуют результатам элюции с использованием 50 мМ фосфата Na/500 мМ NaCl/250 мМ имидазола. Дорожки 14-16 соответствуют результатам элюции с использованием 50 мМ фосфата Na/500 мМ NaCl/500 мМ имидазола.
Количество белка в выделенном элюированном растворе измеряли способом количественного анализа белков и подтверждали с использованием электрофореза в ПААГ с SDS. Как показано на фигуре 33, после подтверждения белок TOM70-(GGGGS)3-UB-RKIP быстро охлаждали жидким азотом и хранили в криогенном морозильном устройстве при -80°C. В этом случае дорожка M на фигуре 33 соответствует маркеру молекулярной массы белков, и на дорожке 1 показан белок TOM70-(GGGGS)3-UB-RKIP, полученный после диализа в буферном растворе PBS.
Пример 4. Получение слитого белка, содержащего PTEN
Пример 4.1. Амплификация гена PTEN
Для экспрессии PTEN человека (гомолога фосфатазы и тензина) в рекомбинантном белке выделяли тотальную РНК из эпителиальных клеток человека и синтезировали из нее кДНК. Фибробласты (дермальные фибробласты человека) культивировали в среде с 10% сыворотки в условиях 5% диоксида углерода и 37°C (1×106 клетки). Затем РНК получали тем же способом, что и в примере 1.1., а затем ее использовали в качестве матрицы для полимеразной цепной реакции гена PTEN.
Для получения из дермальных фибробластов человека гена PTEN, из которого удаляли последовательность сигнального пептида, синтезировали праймер T2PTEN, кодирующий с амино-концевого треонина, и праймер XPTEN(noT), кодирующий с карбокси-конца, а затем осуществляли ПЦР с использованием кДНК, полученной, как описано выше, в качестве матрицы. Последовательность каждого праймера приведена в таблице 10 ниже.
[Таблица 10]
кДНК, полученную, как описано выше, использовали в качестве матрицы, и смешивали 0,2 пмоль праймера T2PTEN и 0,2 пмоль праймера XPTEN(noT) с 0,2 нМ dNTP, 1-кратным реакционным буферным раствором для ДНК-полимеразы AccuPrime Taq (Invitrogen, USA) и 1 ед. ДНК-полимеразы AccuPrime Taq. Затем в устройстве для полимеразной цепной реакции осуществляли реакции амплификации при 95°C в течение 40 секунд, 58°C в течение 30 секунд, 72°C в течение 1 минуты в течение 40 циклов. После реакции амплифицированный фрагмент ДНК размером приблизительно 1200 п.н. выделяли посредством электрофореза в 1% агарозном геле, а затем встраивали в вектор pGEM-T easy (Promega, USA) с использованием ДНК-лигазы T4. В результате секвенирования полученной таким образом ДНК подтверждали получение кДНК, кодирующей белок RKIP человека. Полученный ген PTEN обозначали как pTA-PTEN (фигура 34), и последовательность оснований PTEN представлена последовательностью оснований SEQ ID NO: 35.
Пример 4.2. Получение экспрессирующего вектора E. coli для белка PTEN
Пример 4.2.1. Получение плазмиды pET11c-TOM70-(GGGGS)3-UB-PTEN
Для получения белка PTEN в форме, с которой слиты TOM70, связывающийся с митохондриальной внешней мембраной, линкер (GGGGSGGGGSGGGGS) и убиквитин, получали экспрессирующий вектор, способный экспрессировать ген PTEN в форме, с которой слиты TOM70, линкер и убиквитин.
Плазмиду pTA-PTEN, полученную в примере 4.1. выше, расщепляли с помощью ферментов рестрикции SacII и XhoI, и получали фрагмент ДНК размером приблизительно 1200 п.н. посредством электрофореза в 2% агарозном геле. Затем его встраивали в вектор pET11c-TOM70-(GGGGS)3-UB-(p53), расщепленный с помощью ферментов рестрикции SacII и XhoI, с использованием ДНК-лигазы T4 для получения плазмиды pET11c-TOM70-(GGGGS)3-UB-PTEN(фигура 35). В этом случае TOM70-(GGGGS)3-UB-PTEN представлен последовательностью оснований SEQ ID NO: 36.
Штамм E. coli BL21(DE3) трансформировали с использованием плазмиды pET11c-TOM70-(GGGGS)3-UB-PTEN. Затем трансформированный штамм культивировали в твердой среде Лурия-Бертани (LB), в которую добавляли антибиотик ампициллин, а затем полученные колонии культивировали в жидкой среде LB при 37°C. Затем, когда плотность клеток достигала оптической плотности приблизительно 0,2 при OD600, добавляли IPTG для достижения конечной концентрации 0,5 мМ, а затем осуществляли культивирование при встряхивании еще в течение приблизительно 4 часов.
Часть клеток E. coli получали посредством центрифугирования, а затем клетки разрушали и осуществляли электрофорез в ПААГ с SDS. Как показано на фигуре 36, подтверждали экспрессию белка PTEN, имеющего размер приблизительно 73 кДа, в форме, с которой слиты TOM70, линкер и убиквитин. В этом случае дорожка M на фигуре 36 соответствует маркеру молекулярной массы белков, дорожка 1 соответствует преципитату, центрифугируемому после разрушения E. coli через 4 часа после добавления IPTG, и дорожка 2 соответствует супернатанту, центрифугируемому после разрушения E. coli.
Пример 4.3 Выделение и очистка рекомбинантного белка TOM70-(GGGGS)3-UB-PTEN, полученного из E. coli
Белок TOM70-(GGGGS)3-UB-PTEN выделяли и очищали тем же способом, что и в примере 1.3.1. В результате элюировали белок TOM70-(GGGGS)3-UB-PTEN (фигура 37). В этом случае дорожка M на фигуре 37 соответствует маркеру молекулярной массы белков, и дорожка 1 соответствует нагруженному образцу для никель-аффинной хроматографии. На дорожке 2 видно, что он не связан с никель-аффинной смолой. Дорожка 3 соответствует результатам элюции с использованием раствора 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/10 мМ имидазола. Дорожка 4 соответствует результатам элюции с использованием раствора 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/50 мМ имидазола. Дорожки 5-8 соответствуют результатам элюции с использованием раствора 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/100 мМ имидазола. Дорожки 9-10 соответствуют результатам элюции с использованием раствора 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/250 мМ имидазола. Дорожка 11 соответствует результатам элюции с использованием раствора 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/500 мМ имидазола.
Количество белка в выделенном элюированном растворе измеряли способом количественного анализа белков и подтверждали с использованием электрофореза в ПААГ с SDS. Как показано на фигуре 38, после подтверждения белок TOM70-(GGGGS)3-UB-PTEN быстро охлаждали жидким азотом и хранили в криогенном морозильном устройстве при -80°C. В этом случае дорожка M на фигуре 38 соответствует маркеру молекулярной массы белков, и на дорожке 1 показан белок TOM70-(GGGGS)3-UB-PTEN, полученный после диализа в буферном растворе PBS.
Пример 5. Получение слитого белка, содержащего белок митохондриальной внешней мембраны, убиквитин и GFP
Пример 5.1. Выделение и очистка рекомбинантного белка UB-GFP-TOM7, полученного из E. coli
Производственный штамм E. coli BL21 (DE3), экспрессирующий белок UB-GFP-TOM7 в зрелой форме, с которой слит убиквитин, инокулировали в жидкую среду LB, и культивировали при 37°C. Когда оптическая плотность достигала 0,3 при OD600, культуру помещали в холодильную камеру для снижения температуры культурального раствора, и температуру инкубатора изменяли на 18°C, а затем добавляли 0,5 мМ IPTG и осуществляли культивирование при встряхивании еще в течение 1 дня для экспрессии белка GFP-TOM7 в зрелой форме, с которой слит убиквитин.
После завершения культивирования клетки выделяли посредством центрифугирования, и выделенные клетки однократно промывали с использованием PBS, а затем клетки суспендировали с использованием раствора 50 мМ фосфата натрия, 500 мМ NaCl, 10 мМ имидазола, pH 8,0, и суспендированные клетки разрушали с использованием ультразвукового гомогенизатора. Разрушенные клетки центрифугировали с использованием высокоскоростной центрифуги, а затем выделяли супернатант, и выделенный супернатант фильтровали с использованием фильтра 0,45 мкм, а затем нагружали на упакованную никелевую колонку для хроматографии для осуществления первичной очистки.
Нагружали раствор для разрушения клеток, содержащий белок UB-GFP-TOM7 в зрелой форме, с которой слит убиквитин, а затем пропускали промывочный раствор 50 мМ фосфата натрия, 500 мМ NaCl, 20 мМ имидазола, pH 8,0, пока несвязанные примеси не будут неопределимыми, и элюировали белок в соответствии с градиентом концентрации с использованием раствора 50 мМ фосфата натрия, 500 мМ NaCl, 500 мМ имидазола, pH 8,0 (фигура 39). В этом случае дорожка M на фигуре 39 соответствует маркеру молекулярной массы белков, и дорожка 1 соответствует нагруженному образцу для никель-аффинной хроматографии. На дорожке 2 видно, что он не связан с никель-аффинной смолой. Дорожка 3 соответствует результатам элюции с использованием 50 мМ фосфата Na/500 мМ NaCl/20 мМ имидазола. Дорожка 4 соответствует результатам элюции с использованием 50 мМ фосфата Na/500 мМ NaCl/55 мМ имидазола. Дорожка 5 соответствует результатам элюции с использованием 50 мМ фосфата Na/500 мМ NaCl/60 мМ имидазола. Дорожка 6 соответствует результатам элюции с использованием 50 мМ фосфата Na/500 мМ NaCl/65 мМ имидазола. Дорожка 7 соответствует результатам элюции с использованием 50 мМ фосфата Na/500 мМ NaCl/70 мМ имидазола. Дорожка 8 соответствует результатам элюции с использованием 50 мМ фосфата Na/500 мМ NaCl/75 мМ имидазола. Дорожка 9 соответствует результатам элюции с использованием 50 мМ фосфата Na/500 мМ NaCl/80 мМ имидазола. Дорожка 10 соответствует результатам элюции с использованием 50 мМ фосфата Na/500 мМ NaCl/85 мМ имидазола. Дорожка 11 соответствует результатам элюции с использованием 50 мМ фосфата Na/500 мМ NaCl/90 мМ имидазола. Дорожка 12 соответствует результатам элюции с использованием 50 мМ фосфата Na/500 мМ NaCl/95 мМ имидазола. Дорожка 13 соответствует результатам элюции с использованием 50 мМ фосфата Na/500 мМ NaCl/100 мМ имидазола. Дорожка 14 соответствует результатам элюции с использованием 50 мМ фосфата Na/500 мМ NaCl/105 мМ имидазола.
Для удаления имидазола из элюируемого раствора осуществляли диализ с использованием принципа осмотического давления с помощью раствора 50 мМ фосфата натрия, 500 мМ NaCl, pH 8,0 (фигура 40). Идентифицированный конечный UB-GFP-TOM7 быстро охлаждали жидким азотом и хранили в криогенном морозильном устройстве при -80°C. В этом случае дорожка M на фигуре 40 соответствует маркеру молекулярной массы белков, и дорожка 1 соответствует белку, полученному после диализа, осуществленного с помощью раствора 50 мМ фосфата Na/500 мМ NaCl после смешивания с фракцией слитого белка.
Пример 5.2. Выделение и очистка рекомбинантного белка TOM70-(GGGGS)3-UB-GFP, полученного из E. coli
Производственный штамм E. coli BL21 (DE3), экспрессирующий рекомбинантный белок TOM70-(GGGGS)3-UB-GFP, инокулировали в жидкую среду LB и культивировали при 37°C. Когда оптическая плотность достигала 0,3 при OD600, культуру помещали в холодильную камеру для снижения температуры культурального раствора и температуру инкубатора изменяли на 18°C, а затем добавляли 0,5 мМ IPTG и осуществляли культивирование при встряхивании еще в течение 1 дня для экспрессии рекомбинантного белка TOM70-(GGGGS)3-UB-GFP.
После завершения культивирования клетки выделяли посредством центрифугирования и выделенные клетки промывали однократно с использованием PBS, а затем клетки суспендировали с использованием раствора 50 мМ фосфата натрия, 500 мМ NaCl, 10 мМ имидазола, pH 8,0, и суспендированные клетки разрушали с использованием ультразвукового гомогенизатора. Разрушенные клетки центрифугировали с использованием высокоскоростной центрифуги, а затем выделяли супернатант и выделенный супернатант фильтровали с использованием фильтра 0,45 мкм, а затем нагружали на упакованную никелевую колонку для хроматографии для осуществления первичной очистки.
Раствор после разрушения клеток, содержащий рекомбинантный белок TOM70-(GGGGS)3-UB-GFP, нагружали на колонку, содержащую никелевые смолы, а затем, пока несвязанные примеси не будут неопределимыми, пропускали промывочный раствор 50 мМ фосфата натрия, 500 мМ NaCl, 20 мМ имидазола, pH 8,0. Затем белок элюировали с использованием раствора 50 мМ фосфата натрия, 500 мМ NaCl, 500 мМ имидазола, pH 8,0, заменяя концентрацию имидазола на 50 мМ, 100 мМ, 250 мМ, 500 мМ (фигура 41). В этом случае дорожка M на фигуре 41 соответствует маркеру молекулярной массы белков, и дорожка 1 соответствует нагруженному образцу для никель-аффинной хроматографии. На дорожке 2 видно, что он не связан с никель-аффинной смолой. Дорожка 3 соответствует результатам элюции с использованием 50 мМ фосфата Na/500 мМ NaCl/20 мМ имидазола. Дорожка 4 соответствует результатам элюции с использованием 50 мМ фосфата Na/500 мМ NaCl/50 мМ имидазола. Дорожки 5-8 соответствуют результатам элюции с использованием 50 мМ фосфата Na/500 мМ NaCl/100 мМ имидазола. Дорожки 9-11 соответствуют результатам элюции с использованием 50 мМ фосфата Na/500 мМ NaCl/250 мМ имидазола. Дорожка 12 соответствует результатам элюции с использованием 50 мМ фосфата Na/500 мМ NaCl/500 мМ имидазола.
Элюированный раствор, полученный посредством хроматографии на никеле, подвергали замене буфера буферным раствором PBS с использованием принципа осмотического давления. После завершения замены буфера выделенный конечный белок TOM70-(GGGGS)3-UB-GFP идентифицировали с использованием количественного анализа и электрофореза в ПААГ с SDS. Как показано на фигуре 42, после завершения идентификации белок TOM70-(GGGGS)3-UB-GFP быстро охлаждали жидким азотом и хранили в криогенном морозильном устройстве при -80°C. В этом случае дорожка M на фигуре 42 соответствует маркеру молекулярной массы белков, и дорожка 1 соответствует белку TOM70-(GGGGS)3-UB-GFP, полученному после диализа, осуществленного в буферном растворе PBS.
II. Получение слитого белка, содержащего белок для таргетинга митохондриальной внешней мембраны и белок для таргетинга мишени
Пример 6. Получение слитого белка, содержащего scFvHER2
Пример 6.1. Синтез гена scFvHER2
В случае экспрессии scFvHER2 человека в рекомбинантном белке ген scFvHER2, полученный посредством синтеза гена на заказ в Bionics Co., Ltd., обозначали как pUC57-scFvHER2, и последовательность оснований scFvHER2 являлась такой же, как и последовательность оснований SEQ ID NO: 37.
Пример 6.2. Получение экспрессирующего вектора для белка scFvHER2
Пример 6.2.1. pET15b-UB-scFvHER2-TOM7
Для получения белка scFvHER2 в форме, с которой слиты убиквитин и TOM7, связывающийся с митохондриальной внешней мембраной, получали экспрессирующий вектор, способный экспрессировать ген scFvHER2 в форме, с которой слиты убиквитин и TOM7.
Плазмиду pUC57-scFvHER2, полученную в примере 6.1., расщепляли с помощью ферментов рестрикции SacII и XhoI и получали фрагмент ДНК размером приблизительно 750 п.н. посредством электрофореза в 2% агарозном геле, а затем его встраивали в вектор pET15b-UB-(p53)-TOM7, расщепленный с помощью ферментов рестрикции SacII и XhoI, с использованием ДНК-лигазы T4 для получения плазмиды pET15b-UB-scFvHER2-TOM7 (фигура 39). В этом случае UB-scFvHER2-TOM7 представлен последовательностью оснований SEQ ID NO: 38.
Штамм E. coli BL21(DE3) трансформировали с использованием плазмиды pET15b-UB-scFvHER2-TOM7. Затем трансформированный штамм культивировали в твердой среде Лурия-Бертани (LB), в которую добавляли антибиотик ампициллин, а затем полученные колонии культивировали в жидкой среде LB при 37°C. Затем, когда плотность клеток достигала оптической плотности приблизительно 0,2 при OD600, добавляли IPTG для достижения конечной концентрации 1 мМ, а затем осуществляли культивирование при встряхивании еще в течение приблизительно 4 часов.
Часть клеток E. coli получали посредством центрифугирования, а затем клетки разрушали и осуществляли электрофорез в ПААГ с SDS. Как показано на фигуре 44, подтверждали, что экспрессировали белок scFvHER2, имеющий размер приблизительно 35 кДа, в форме, с которой слиты убиквитин и TOM7. В этом случае дорожка M на фигуре 44 соответствует маркеру молекулярной массы белков, дорожка 1 соответствует преципитату, центрифугируемому после разрушения E. coli через 4 часа после добавления IPTG, и дорожка 2 соответствует супернатант центрифугируемому после разрушения E. coli.
Пример 6.2.2. Получение pCMV-scFvHER2-TOM7-myc/His
Для получения белка scFvHER2 в форме, с которой слит TOM7, связывающийся с митохондриальной внешней мембраной, получали экспрессирующий вектор для клеток животных, способный экспрессировать scFvHER2 в форме, с которой слит TOM7. Для получения генов TOM7 и scFvHER2 получали праймер RscFvHER2 и праймер XTOM7(noT). Последовательность каждого праймера приведена в таблице 11 ниже.
[Таблица 11]
Плазмиду pET15b-UB-scFvHER2-TOM7, полученную в примере 6.2.1., использовали в качестве матрицы и смешивали 0,2 пмоль праймера (RscFvHER2) и 0,2 пмоль праймера (XTOM7(noT)) с 0,2 нМ dNTP, 1-кратным реакционным буферным раствором для ДНК-полимеразы AccuPrime Taq (Invitrogen, USA) и 1 ед. ДНК-полимеразы AccuPrime Taq. Затем для получения гена scFvHER2-TOM7 в устройстве для полимеразной цепной реакции осуществляли реакции амплификации при 95°C в течение 40 секунд, 58°C в течение 30 секунд, 72°C в течение 1 минуты в течение 25 циклов. Амплифицированный ген scFvHER2-TOM7 расщепляли с помощью ферментов рестрикции EcoRI и XhoI и получали фрагменты ДНК размером приблизительно 850 п.н., соответственно, посредством электрофореза в 1% агарозном геле, а затем встраивали их в вектор pcDNA3.1-myc/His A, расщепленный с помощью ферментов рестрикции EcoRI и XhoI, с использованием ДНК-лигазы T4 для получения плазмиды pCMV-scFvHER2-TOM7-myc/His (фигура 45).
В этом случае scFvHER2-TOM7-myc/His представлен последовательностью оснований SEQ ID NO: 41. С помощью него трансфицировали клетки животных CHO с использованием плазмиды pCMV-scFvHER2-TOM7-myc/His, клетки разрушали, а затем осуществляли электрофорез в ПААГ с SDS и анализировали посредством вестерн-блоттинга с использованием антитела против c-myc. Как показано на фигуре 46, подтверждали экспрессию белка scFvHER2, имеющего размер приблизительно 35 кДа, в форме, с которой слит TOM7. В этом случае дорожка M на фигуре 46 соответствует маркеру молекулярной массы белков, и на дорожке 1 видно, что с помощью него трансфицировали клетки животных CHO, клетки разрушали, а затем осуществляли электрофорез в ПААГ с SDS, а затем подтверждали посредством вестерн-блоттинга с использованием антитела против c-myc.
Пример 6.3.Выделение и очистка рекомбинантного белка UB-ScFvHER2-TOM7, полученного из E. coli
Белок UB-ScFvHER2-TOM7 выделяли и очищали тем же способом, что и в примере 1.3.1. В результате элюировали белок UB-ScFvHER2-TOM7 (фигура 47). В этом случае дорожка M на фигуре 47 соответствует маркеру молекулярной массы белков, и дорожка 1 соответствует нагруженному образцу для никель-аффинной хроматографии. На дорожке 2 видно, что он не связан с никель-аффинной смолой. Дорожка 3 соответствует результатам элюции с использованием 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/10 мМ имидазола. Дорожки 4-5 соответствуют результатам элюции с использованием 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/50 мМ имидазола. Дорожки 6-8 соответствуют результатам элюции с использованием 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/100 мМ имидазола. Дорожки 9-10 соответствуют результатам элюции с использованием 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/250 мМ имидазола. Дорожка 11 соответствует результатам элюции с использованием 8 М мочевины/50 мМ фосфата Na/500 мМ NaCl/500 мМ имидазола.
Количество белка в выделенном элюированном растворе измеряли способом количественного анализа белков и подтверждали с использованием электрофореза в ПААГ с SDS. Как показано на фигуре 48, после подтверждения белок UB-ScFvHER2-TOM7 быстро охлаждали жидким азотом и хранили в криогенном морозильном устройстве при -80°C. В этом случае дорожка M на фигуре 48 соответствует маркеру молекулярной массы белков, и дорожка 1 соответствует белку UB-ScFvHER2-TOM7, полученному после диализа в буферном растворе PBS.
Пример 7. Получение слитого белка, содержащего scFvMEL
Пример 7.1. Синтез гена scFvMEL
Для экспрессии scFvMEL человека в рекомбинантном белке в виде фрагмента антитела против меланомы, ген scFvMEL полученный посредством синтеза генов на заказ в Bionics Co., Ltd., обозначали как pUC57-scFvMEL, и последовательность оснований scFvMEL являлась такой же, как и последовательность оснований SEQ ID NO: 42.
Пример 7.2. Получение экспрессирующего вектора для белка scFvMEL
Пример 7.2.1. Получение pET15b-UB-scFvMEL-TOM7
Для получения белка scFvMEL в форме, с которой слиты убиквитин и TOM7, связывающийся с митохондриальной внешней мембраной, получали экспрессирующий вектор, способный экспрессировать scFvMEL в форме, с которой слиты убиквитин и TOM7.
Плазмиду pUC57-scFvMEL, полученную в примере 7.1., расщепляли с помощью ферментов рестрикции SacII и XhoI и получали фрагмент ДНК размером приблизительно 750 п.н. посредством электрофореза в 2% агарозном геле, а затем встраивали в вектор pET15b-UB-(p53)-TOM7, расщепленный с помощью ферментов рестрикции SacII и XhoI, с использованием ДНК-лигазы T4 для получения плазмиды pET15b-UB-scFvMEL-TOM7 (фигура 49). В этом случае UB-scFvMEL-TOM7 представлен последовательностью оснований SEQ ID NO: 43.
Штамм E. coli BL21(DE3) трансформировали с использованием плазмиды pET15b-UB-scFvMEL-TOM7. Затем трансформированный штамм культивировали в твердой среде Лурия-Бертани (LB), в которую добавляли антибиотик ампициллин, а затем полученные колонии культивировали в жидкой среде LB в инкубаторе-встряхивателе при 37°C. Затем, когда плотность клеток достигала оптической плотности приблизительно 0,2 при OD600, добавляли IPTG для достижения конечной концентрации 1 мМ, а затем осуществляли культивирование при встряхивании еще в течение приблизительно 4 часов.
Часть клеток E. coli получали посредством центрифугирования, а затем клетки разрушали и осуществляли электрофорез в ПААГ с SDS. Как показано на фигуре 50, подтверждали, что экспрессировали белок scFvMEL, имеющий размер приблизительно 35 кДа, в форме, с которой слиты убиквитин и TOM7. В этом случае дорожка M на фигуре 50 соответствует маркеру молекулярной массы белков, дорожка 1 соответствует преципитату, центрифугируемому после разрушения E. coli через 4 часа после добавления IPTG, и дорожка 2 соответствует супернатанту, центрифугируемому после разрушения E. coli.
Пример 7.2.2. Получение pCMV-scFvMEL-TOM7-myc/His
Для получения белка scFvMEL в форме, с которой слит TOM7, связывающийся с митохондриальной внешней мембраной, получали экспрессирующий вектор для клеток животных, способный экспрессировать scFvMEL в форме, с которой слит TOM7. Для получения генов TOM7 и scFvMEL получали праймер (RscFvMEL). Последовательность каждого праймера приведена в таблице 12 ниже.
[Таблица 12]
Плазмиду pET15b-UB-scFvMEL-TOM7, полученную в примере 6.2.1., использовали в качестве матрицы, и смешивали 0,2 пмоль праймера RscFvMEL и 0,2 пмоль праймера XTOM7(noT) с 0,2 нМ dNTP, 1-кратным реакционным буферным раствором для ДНК-полимеразы AccuPrime Taq (Invitrogen, USA) и 1 ед. ДНК-полимеразы AccuPrime Taq. Затем для получения scFvMEL-TOM7 в устройстве для полимеразной цепной реакции осуществляли реакции амплификации при 95°C в течение 40 секунд, 58°C в течение 30 секунд, 72°C в течение 1 минуты в течение 25 циклов. Амплифицированный ген scFvMEL-TOM7 расщепляли с помощью ферментов рестрикции EcoRI и XhoI и получали фрагмент ДНК размером приблизительно 850 п.н. посредством электрофореза в 1% агарозном геле. Затем его встраивали в вектор pcDNA3.1-myc/His A, расщепленный с помощью ферментов рестрикции EcoRI и XhoI, с использованием ДНК-лигазы T4 для получения плазмиды pCMV-scFvMEL-TOM7-myc/His (фигура 51). В этом случае scFvMEL-TOM7-myc/His представлен последовательностью оснований SEQ ID NO: 45.
С помощью него трансфицировали клетки животных CHO с использованием плазмиды pCMV-scFvMEL-TOM7-myc/His, клетки разрушали, а затем осуществляли электрофорез в ПААГ с SDS и анализировали посредством вестерн-блоттинга с использованием антитела против c-myc. Как показано на фигуре 52, подтверждали экспрессию белка scFvMEL, имеющего размер приблизительно 35 кДа, в форме, с которой слит TOM7. В этом случае дорожка M на фигуре 52 соответствует маркеру молекулярной массы белков, и дорожка 1 видно, что с помощью него трансфицировали клетки животных CHO, и клетки разрушали, а затем осуществляли электрофорез в ПААГ с SDS, а затем подтверждали посредством вестерн-блоттинга с использованием антитела против c-myc.
Пример 8. Получение слитого белка, содержащего scFvPD-L1
Пример 8.1. Синтез гена scFvPD-L1
В случае экспрессии scFvPD-L1 человека в рекомбинантном белке ген scFvPD-L1, полученный посредством синтеза генов на заказ в Bionics Co., Ltd., обозначали как pUC57-scFvPD-L1, последовательность оснований которого являлась такой же, как и последовательность оснований SEQ ID NO: 46.
Пример 8.2. Получение экспрессирующего вектора для белка scFvPD-L1
Пример 8.2.1. Получение pCMV-scFvPD-L1-TOM7-myc/His
Для получения белка scFvPD-L1 в форме, с которой слит TOM7, связывающийся с митохондриальной внешней мембраной, получали экспрессирующий вектор для клеток животных, способный экспрессировать scFvPD-L1 в форме, с которой слиты убиквитин и TOM7.
Плазмиду pUC57-scFvPD-L1 расщепляли с помощью ферментов рестрикции EcoRI и XhoI и получали фрагмент ДНК размером приблизительно 760 п.н. посредством электрофореза в 1% агарозном геле. Затем его встраивали в вектор pCMV-(scFvMEL)-TOM7-myc/His, расщепленный с помощью ферментов рестрикции EcoRI и XhoI, с использованием ДНК-лигазы T4 для получения плазмиды pCMV-scFvPD-L1-TOM7-myc/His (фигура 53). В этом случае scFvPD-L1-TOM7-myc/His представлен последовательностью оснований SEQ ID NO: 47.
С помощью него трансфицировали клетки животных CHO с использованием плазмиды pCMV-scFvPD-L1-TOM7-myc/His, и клетки разрушали, а затем осуществляли электрофорез в ПААГ с SDS и анализировали посредством вестерн-блоттинга с использованием антитела против c-myc. Как показано на фигуре 54, подтверждали экспрессию белка scFvPD-L1, имеющего размер приблизительно 35 кДа, в форме, с которой слит TOM7. В этом случае дорожка M на фигуре 54 соответствует маркеру молекулярной массы белков, и дорожка 1 видно, что с помощью него трансфицировали клетки животных CHO, клетки разрушали, а затем осуществляли электрофорез в ПААГ с SDS, а затем подтверждали посредством вестерн-блоттинга с использованием антитела против c-myc.
III. Получение модифицированных митохондрий, с которыми связан слитый белок
Пример 9. Получение модифицированных митохондрий
Следующий эксперимент проводили для подтверждения того, связывается ли флуоресцентный белок, слитый с участком связывания митохондриальной внешней мембраны, с внешней мембраной митохондрий. Сначала митохондрии выделяли из мезенхимальных стволовых клеток, полученных из пуповины (UC-MSC) способом центрифугирования. Затем их окрашивали красителем MitoTracker CMXRos Red. Их смешивали с рекомбинантным белком TOM70-(GGGGS)3-UB-GFP, выделенным из E. coli, как описано выше, и инкубировали при температуре окружающей среды в течение приблизительно 30 минут.
Затем непрореагировавший белок удаляли посредством центрифугирования и дважды промывали буферным раствором PBS. Затем флуоресцентный белок в форме, связанной с митохондриями, наблюдали с использованием флуоресцентного микроскопа. В качестве контрольной группы использовали очищенный белок GFP, не содержащий участок связывания митохондриальной внешней мембраны. В результате подтверждали, что флуоресцентный белок, слитый с участком связывания митохондриальной внешней мембраны (TOM70-(GGGGS)3-UB-GFP), находился в том же месте, что и митохондрии из мезенхимальных стволовых клеток, полученных из пуповины (UC-MSC) (фигура 55a, фигура 55b).
Пример 10. Подтверждение способности рекомбинантного белка p53 связываться с чужеродной митохондриальной внешней мембраной
Митохондрии, выделенные из мезенхимальных стволовых клеток, полученных из пуповины, способом центрифугирования, смешивали с очищенным рекомбинантным белком TOM70-(GGGGS)3-UB-p53 или UB-p53-TOM7 и позволяли им связываться в соотношении 1:1 в условиях реакции при 4°C в течение 1 часа. В качестве контрольной группы использовали митохондрии, не смешанные с белком. Возможность связывания митохондрий и p53 подтверждали способом вестерн-блоттинга (фигура 56).
Сначала связывались митохондрии и белок p53, а затем осуществляли центрифугирование при 13000 об./мин. в течение 10 минут для получения митохондрий или митохондрий, с которыми связан p53, в виде осадка. Белок, несвязанный с митохондриями, удаляли посредством двукратной промывки PBS, и промытый осадок подвергали электрофорезу белков (в ПААГ с SDS), а затем вестерн-блоттингу. В качестве первичного антитела использовали антитело кролика против p53 и в качестве вторичного антитела использовали HRP-конъюгированное антитело против IgG кролика. Подтверждали наличие полос в том же положении, соответствующем размеру 60 кДа, молекулярной массе, ожидаемой в экспериментальной группе для митохондрий, связавшихся с TOM70-(GGGGS)3-UB-p53 или UB-p53-TOM7, по сравнению с контрольной группой митохондрий в отдельности, несвязавшихся с белком (фигура 56).
IV. Подтверждение активности модифицированных митохондрий, с которыми связан активный белок
Пример 11. Выделение и внутриклеточная инъекция чужеродных митохондрий
Митохондрии выделяли из мезенхимальных стволовых клеток, полученных из пуповины (UC-MSC), способом центрифугирования. Выделенные митохондрии окрашивали с помощью красителя Mitotracker CMX Ros и концентрацию и общее количество выделенных митохондрий подтверждали способом количественного анализа BCA, и 0 мкг, 1 мкг, 5 мкг, 10 мкг, 50 мкг, 100 мкг митохондрий инъецировали в клетки SNU-484, линию клеток рака желудка, способом центрифугирования. В результате эксперимента с помощью флуоресцентного микроскопа подтверждали, что количество митохондрий, инъецируемых в клетки, зависело от концентрации митохондрий (фигура 57).
Пример 12. Подтверждение влияния нормальных митохондрий на злокачественные клетки
Следующий эксперимент проводили для исследования того, как митохондрии, полученные из нормальных клеток, влияют на пролиферацию злокачественных клеток и продукцию ROS. Сначала клетки печени (WRL-68), фибробласты и мезенхимальные стволовые клетки, полученные из пуповины (UC-MSC), выбирали в качестве клеток-доноров митохондрий. Митохондрии выделяли из клеток способом фракционирования центрифугированием, соответственно. Злокачественной клеткой, используемой в качестве клетки-реципиента митохондрий, являлась эпидермальная злокачественная клетка кожи, линия клеток A431. В этом случае митохондрии доставляют в эпидермальные злокачественные клетки кожи с использованием центробежной силы в соответствии с концентрацией (см. патентную заявку Кореи № 10-2017-0151526).
Через 24, 48 и 72 часа после введения наблюдали пролиферацию эпидермальных злокачественных клеток кожи и продукцию активных форм кислорода (ROS). В результате подтверждали, что, когда митохондрии, полученные из нормальных клеток различного происхождения, инъецировали в злокачественные клетки, наблюдали эффект ингибирования пролиферации злокачественных клеток в зависимости от концентрации. Кроме того, подтверждали, что продукция ROS в злокачественных клетках подвергалась ингибированию в зависимости от концентрации нормальных митохондрий (фигуры 58 и 59).
Пример 13. Подтверждение влияния нормальных митохондрий на резистентность к лекарственным средствам
Авторы настоящего изобретения исследовали влияние на резистентность к лекарственным средствам, экспрессию генов антиоксидантов, метастазирование злокачественных новообразований, являющиеся признаками злокачественных клеток, когда митохондрии, полученные из нормальных клеток, инъецировали в злокачественные клетки, следующими способами. Сначала нормальные клетки печени (WRL-68) использовали в качестве клеток-доноров митохондрий, митохондрии выделяли из клеток способом фракционирования центрифугированием и использовали митохондрии. Клетки HepG2, линию злокачественных клеток печени, использовали в качестве злокачественных клеток-реципиентов митохондрий. Митохондрии доставляли в злокачественные клетки печени с использованием центробежной силы в соответствии с концентрацией, а затем подтверждали, что при наблюдении резистентности к противоопухолевому средству доксорубицину линии злокачественных клеток, в которые вводили митохондрии, демонстрировали более высокую чувствительность к лекарственным средствам (фигура 60).
Пример 14. Подтверждение влияния нормальных митохондрий на антиоксидантный эффект
Т.к. митохондрии, выделенные из нормальных клеток, инъецировали в клетки HepG2, линию злокачественных клеток печени, в соответствии с концентрацией, подтверждали повышение экспрессии генов фермента каталазы, антиоксидантного белка, и SOD-2 (супероксиддисмутазы-2) в злокачественных клетках (фигура 61).
Пример 15. Подтверждение влияния нормальных митохондрий на метастазирование злокачественных клеток
Что касается метастазирования, подтверждали наличие экспрессии гена α-актина гладких мышц (α-SMA), одного из генов, участвующих в EMT (эпителиально-мезенхимальном переходе). В этом случае обнаружено, что в случае злокачественных клеток печени, в которые вводили митохондрии, экспрессия белка α-SMA значительно снижалась в зависимости от концентрации митохондрий по сравнению со злокачественными клетками печени, в которые не вводили митохондрии. В отличие от этого, обнаружено, что белок E-кадгерин, один из белков клеточной адгезии, повышался в зависимости от концентрации митохондрий (фигура 62). Подтверждали, что изменения белков, которые, как известно, участвуют в метастазировании злокачественных новообразований, происходили под действием нормальных митохондрий, инъецированных в злокачественные клетки, и, таким образом, также влияли на метастазирование злокачественных клеток.
Пример 16. Подтверждение нагрузки рекомбинантного белка p53 на чужеродной митохондриальной внешней мембране и инъекции в клетки
Митохондрии выделяли из мезенхимальных стволовых клеток, полученных из пуповины, способом центрифугирования, а затем окрашивали с помощью красителя Mitotracker CMX Ros, смешивали с очищенным рекомбинантным белком TOM70-(GGGGS)3-UB-p53 или UB-p53-TOM7 и инкубировали в соотношении 1:1 в условиях реакции при 4°C в течение 1 часа, а затем центрифугировали для удаления непрореагировавших белков, а затем дважды промывали буферным раствором PBS, а затем митохондрии в форме, с которой связан белок p53, инъецировали в клетки SNU-484, линию клеток рака желудка, способом центрифугирования (фигура 63). В этом случае контрольной группой являлась группа, в которой не использовали митохондрии, и группа, в которой использовали митохондрии в отдельности. После одного дня культивирования белок p53, нагруженный на чужеродные митохондрии, инъецированные в клетки, анализировали с помощью флуоресцентного микроскопа с использованием иммуноцитохимии (ICC).
В качестве первичного антитела использовали антитело кролика против p53 и в качестве вторичного антитела использовали конъюгированное с Alexa Fluor 488 антитело козы против IgG кролика. В результате подтверждали, что белок TOM70-(GGGGS)3-UB-p53 (окрашенный зеленым) или UB-p53-TOM7 (окрашенный зеленым), нагруженный на чужеродные митохондрии (окрашенные красным), находился в цитоплазме в клетках, в которые инъецировали чужеродные митохондрии (фигура 64, 200-кратное увеличение, и фигура 65, 400-кратное увеличение). В результате обнаружено, что рекомбинантный белок легко инъецировали в клетку с помощью митохондрий.
Пример 17. Подтверждение активности митохондрий с нагруженным p53 в линии злокачественных клеток
Пример 17.1. Подтверждение апоптотической способности чужеродных митохондрий с нагруженным p53, инъецированных в клетки, с использованием линии клеток рака желудка
Митохондрии, выделенные из мезенхимальных стволовых клеток, полученных из пуповины, способом центрифугирования, смешивали с рекомбинантным белком TOM70-(GGGGS)3-UB-p53 или UB-p53-TOM7, выделенным из E. coli, и им позволяли связываться в соотношении 1:1 в условиях реакции при 4°C в течение 1 часа. В качестве контрольной группы использовали белок UB-p53, несодержащий TOM70, и TOM70-(GGGGS)3-p53, несодержащий убиквитин. Несвязанные белки удаляли посредством центрифугирования и промывки PBS, и митохондрии, с которыми связаны белки, инъецировали в линию клеток рака желудка SNU-484, в которых отсутствуют возможности p53 из-за изменения гена p53, посредством центрифугирования (фигура 66). После одного дня культивирования осуществляли фиксацию 4% параформальдегидом в течение 1 часа, а затем вызывали пермеабилизацию клеток с использованием раствора для пермеабилизации (0,1% буфер цитрата натрия, содержащий 0,1% Triton-X-100, pH 7,4) и проводили реакцию с раствором TUNEL (набор для детекции гибели клеток in situ, TMR RED, Roche) при 37°C в течение 1 часа.
В способе анализа TUNEL часть, в которой происходит фрагментация нуклеиновых кислот (фрагментация ДНК), окрашивается красным, что свидетельствует о том, что происходит апоптоз. В клетках, в которые инъецировали митохондрии, с которыми связан TOM70-(GGGGS)3-ub-p53 или p53-TOM7, обнаруживали значительное красное окрашивание в отличие от контрольной группы, что свидетельствует о том, что происходит апоптоз под действием митохондрий, с которыми связан TOM70-(GGGGS)3-UB-p53 или UB-p53-TOM7. В частности, подтверждали, что в большей степени апоптоз происходил в случае митохондрий, с которыми связан белок в форме TOM70-(GGGGS)3-UB-p53 (фигура 67a).
Пример 17.2. Подтверждение апоптотической способности чужеродных митохондрии с нагруженным p53, с которыми связана люцифераза
Для подтверждения того, сохранялась ли биологическая активность введенного белка TOM70-(GGGGS)3-UB-p53 в клетке-реципиенте после доставки белка TOM70-(GGGGS)3-UB-p53 в форме, связанной с митохондриями, полученными в примере 5.2. выше в клетки-реципиенты, осуществляли клеточный анализ с использованием репортерного гена. Т.к. белок p53 является фактором транскрипции, синтезировали ген, в котором 6 раз повторяется последовательность оснований RRRCWWGYYY (где R представляет собой G или A, W представляет собой A или T, и Y представляет собой C или T), с которой может связываться фактор транскрипции p53, со следующей последовательностью. Последовательность оснований P53-promter-S является следующей (5'-GGG CAT GCT CGG GCA TGC CCG GGC ATG CTC GGG CAT GCC CGG GCA TGC TCG GGC ATG CCC-3') (SEQ ID NO: 91), и последовательность оснований P53-promter-AS является следующей (5'-GGG CAT GCC CGA GCA TGC CCG GGC ATG CCC GAG CAT GCC CGG GCA TGC CCG AGC ATG CCC-3') (SEQ ID NO: 92).
5 мкг синтезированного гена P53-promter-S и 5 мкг синтезированного гена P53-promter-AS инкубировали при 70°C в течение 20 минут, чтобы способствовать синтезу двойной спирали гена, а затем индуцировали реакцию фосфорилирования с использованием фермента полинуклеотид T4-киназы. Двойную спираль гена, в которой индуцировали фосфорилирование, встраивали в вектор pGL3, расщепленный ферментом рестрикции Sma I, и гену, в котором 6 раз повторяется последовательность оснований (RRRCWWGYYY), с которой может связываться фактор транскрипции p53, позволяли связываться с люциферазой, репортерным геном, для получения плазмиды p6xp53-Luc. С помощью плазмиды p6xp53-Luc и плазмиды pRSVb-gal, экспрессирующего вектора бета-галактозидазы, трансформировали клетки HEK293, клетки почки человека, способом с использованием липофектамина.
Затем, через 6 часов, клетки HEK293 обрабатывали комбинацией, в которой 10 мкг митохондрий и 5 мкг, 10 мкг и 20 мкг белка TOM70-(GGGGS)3-UB-p53 связаны, соответственно. В этом случае в качестве контрольной группы клетки обрабатывали 10 мкг митохондрий, с которыми связаны PBS или белок p53, соответственно. Обработанные клетки культивировали в течение 18 часов, а затем измеряли и анализировали активность люциферазы. В этом случае для коррекции эффективности трансформации в качестве скорректированного значения люциферазы определяли значение люциферазы, разделенное на значение, полученное посредством измерения активности бета-галактозидазы.
Подтверждали, что значение люциферазы повышалось в клетках, обработанных комбинацией, в которой связаны 10 мкг митохондрий и 5 мкг, 10 мкг и 20 мкг белка TOM70-(GGGGS)3-UB-p53, соответственно. Таким образом, подтверждали, что белок p53 проникал в клетки и проявлял активность (фигура 67b).
Пример 18. Подтверждение способности чужеродных митохондрий с нагруженным RKIP, инъецированных в клетки, снижать метастазирование линий злокачественных клеток
Митохондрии, выделенные из мезенхимальных стволовых клеток, полученных из пуповины, способом центрифугирования, смешивали с очищенным рекомбинантным белком TOM70-(GGGGS)3-UB-RKIP и позволяли им связываться в соотношении 1:1 в условиях реакции при 4°C в течение 1 часа. Митохондрии, с которыми связан белок, инъецировали посредством центрифугирования в линию злокачественных клеток молочной железы MDA-MB-231, которая, как известно, имеет повышенную способность к метастазированию из-за снижения белка RKIP.
Для подтверждения способности злокачественных клеток к метастазированию осуществляли анализ клеточной инвазии с использованием планшета Transwell. Верхнюю камеру Transwell, имеющую размер пор 8 мкм, покрывали матригелем в течение 30 минут при 37°C. В качестве тестовой группы использовали клетки MDA-MB-231, в которые инъецировали митохондрии в отдельности, и клетки MDA-MB-231, в которые инъецировали митохондрии, с которыми связан белок RKIP. Каждый тип клеток в количестве 1×105 клеток помещали в верхнюю камеру Transwell, содержащую бессывороточную среду, а в нижнюю камеру помещали среду, содержащую 10% бычьей сыворотки. После культивирования при 37°C в течение 12 часов осуществляли фиксацию 4% параформальдегидом в течение 1 часа, а затем клетки, проходящие через матригель, окрашивали 1% кристаллвиолетом.
В результате наблюдения с помощью микроскопа клетки, окрашенные фиолетовым, определяли в мембране ниже верхней камеры, и можно сказать, что это представляет собой процесс, посредством которого происходит метастазирование клеток. Подтверждали, что количество клеток, окрашенных фиолетовым, снижалось в экспериментальной группе, обработанной митохондриями в отдельности, и экспериментальной группе, обработанной митохондриями, с которыми связан RKIP, по сравнению с контрольной группой, которую ничем не обрабатывали. Случайным образом выбирали четыре части, а затем измеряли количество окрашенных клеток и строили график (фигура 68).
IV. Подтверждение скорости доставки модифицированных митохондрий, с которыми связан белок для таргетинга мишени
Пример 19. Подтверждение внутриклеточной экспрессии одноцепочечного вариабельного фрагмента (ScFv) антитела для таргетинга злокачественных клеток и подтверждение связывания с митохондриями в клетках
Для экспрессии pCMV-ScFv-HER2-TOM7 или pCMV-ScFv-MEL-TOM7 или pCMV-ScFv-PD-L1-TOM7 в клетках животного, клетки CHO трансфицировали с помощью ДНК с использованием липофектамина LTX и PLUS или липофектамина 2000. В качестве контрольной группы использовали ДНК GFP-TOM7. Для подтверждения того, что они экспрессировались в клетке и связывались в митохондриях в той же клетке, цитозоль и митохондрии выделяли из трансфицированных клеток способом центрифугирования и корректировали по количеству того же белка с использованием анализа BCA, а затем осуществляли электрофорез в ПААГ, а затем получали результаты посредством вестерн-блоттинга. В качестве первичного антитела использовали моноклональное антитело против c-myc и в качестве вторичного антитела использовали HRP-конъюгированное антитело против IgG мыши.
Полосы белков ScFv-HER2-TOM7 или ScFv-MEL-TOM7 идентифицировали в ожидаемой области 35 кДа. С учетом того, что все они были идентифицированы в митохондриальном слое, можно ожидать, что трансфицированные и экспрессирующиеся белки связаны с митохондриями в клетках посредством TOM7 (фигура 69).
Затем для подтверждения связывания белка-мишени, экспрессирующегося в клетке, с митохондриями в той же клетке, белок ScFv-HER2-TOM7, ScFv-MEL-TOM7 или ScFv-PD-L1-TOM7, экспрессирующийся в клетке, наблюдали с помощью флуоресцентного микроскопа способом иммуноцитохимии (ICC). В качестве первичного антитела использовали моноклональное антитело против c-myc и в качестве вторичного антитела использовали конъюгированное с Alexa Fluor 488 антитело козы против IgG мыши. Митохондрии в клетке окрашивали с помощью красителя Mitotracker CMX Ros. В результате подтверждали, что экспрессирующиеся белки ScFv-HER2-TOM7, ScFv-MEL-TOM7 или ScFv-PD-L1-TOM7 колокализовались с митохондриями и были связаны с митохондриями в клетке (фигуры 70 и 71).
Пример 20. Выделение митохондрий, с которыми связан одноцепочечный вариабельный фрагмент антитела для таргетинга злокачественных клеток, и сравнение инъекции митохондрий в линии клеток рака желудка
Митохондрии выделяли из клеток CHO, которые трансфицировали с помощью pCMV-ScFv-HER2-TOM7 или pCMV-ScFv-PD-L1-TOM7. В качестве контрольной группы выделяли и использовали митохондрии из клеток CHO, которые не трансформировали. Митохондрии, выделенные из каждой клетки, окрашивали с помощью Mitotracker CMX Ros. SNU-484, линию клеток рака желудка, обрабатывали тем же количеством митохондрий и на следующий день с использованием флуоресцентного микроскопа сравнивали и подтверждали количество митохондрий, инъецированных в клетки. Подтверждали, что, по сравнению с контрольной группой, митохондрии, с которыми связан ScFv-HER2-TOM7 или ScFv-PD-L1-TOM7, инъецировали в злокачественные клетки в большем количестве, чем митохондрии, полученные из контрольной группы (фигура 72). Таким образом, обнаружено, что митохондрии, с которыми связан белок-мишень, легче инъецировались в злокачественные клетки, чем при использовании митохондрий в отдельности.
VI. Подтверждение активности in vivo модифицированных митохондрий, с которыми связан активный белок
Пример 21. Конструирование модели ксенотрансплантата (SNU-484) и введение тестируемого вещества
Пример 21.1. Получение злокачественных клеток
В день эксперимента подготавливали линию клеток SNU-484, линию клеток рака желудка, в количестве 5×106 клеток на мышь. Удаляли среду для культивирования клеток, а затем добавляли PBS для промывки клеток. Клетки подвергали диссоциации с использованием раствора трипсин-ЭДТА, а затем клетки помещали в пробирку 50 мл и дважды промывали буферным раствором PBS, а затем добавляли 20 мл PBS и измеряли количество клеток и жизнеспособности. С учетом измеренного количества клеток, количество клеток доводили до 5×106 клеток на мышь, и клетки подготавливали, разделяя их на группы. Объем, подлежащий трансплантации, на одну мышь доводили до того же количества 100 мкл. В качестве контрольной группы подготавливали группу 100 мкл злокачественных клеток в отдельности.
Пример 21.2. Получение тестируемого вещества
Митохондрии, выделенные из мезенхимальных стволовых клеток пуповинной крови, как описано выше, подготавливали для трансплантации в количестве 50 мкг на мышь с учетом концентрации белка. В случае группы, в которой вводили митохондрии в отдельности, митохондрии подготавливали посредством тщательного смешивания с 100 мкл PBS, в котором смешивали злокачественные клетки. В случае группы модифицированных митохондрий, белок TOM70-(GGGGS)3-UB-p53 смешивали в соотношении концентрации 1:1 с количеством митохондрий, подготовленных в пробирке Eppendorf перед смешиванием со злокачественными клетками, и оставляли при температуре окружающей среды на 1 час. По истечении времени реакции супернатант удаляли после центрифугирования при 20000×g в течение 10 минут и получали осадок митохондрий (MT+TOM70-(GGGGS)3-UB-p53), с которыми связан белок. Его дважды промывали буферным раствором PBS, а затем митохондрии (MT+TOM70-(GGGGS)3-UB-p53), с которыми связан белок p53, подготавливали посредством тщательного смешивания с 100 мкл PBS, в котором смешивали злокачественные клетки.
Пример 21.3. Получение экспериментального животного и трансплантация тестируемого вещества
Для трансплантации в образец, подготовленный для групп, добавляли матригель (BD) в том же количестве, что и PBS, и осторожно смешивали с клетками для получения 200 мкл тестируемого вещества на мышь. В этом случае все действия осуществляли на льду. Для конструирования модели мышей Balb/c nude (самок возрастом 7 недель) приобретали в RAONBIO и анестезировали посредством ингаляции изофлурана для трансплантации злокачественных клеток, а затем правую заднюю область (с учетом животного) стерилизовали с помощью тампона, пропитанного спиртом. Затем 200 мкл вводили подкожно в правую заднюю область экспериментального животного с использованием шприца 1 мл, содержащего инъекционный раствор. После введения массу животного и размер опухоли измеряли дважды в неделю и осуществляли анализ результатов при наблюдении в течение до 3 недель (фигура 73).
Пример 21.4. Подтверждение образования опухоли
Объем опухоли вычисляли, измеряя размер по длинной оси и размер по короткой оси опухоли и используя следующее уравнение.
<Математическое уравнение 1>
Длинная ось × короткая ось × короткая ось × 0,5=объем опухоли (мм3)
Пример 21.5. Наблюдение физиологических и морфологических изменений
Для наблюдения физиологических и морфологических изменений у мышей при введении противоопухолевых средств-кандидатов измеряли изменения массы тела и размер опухоли дважды в неделю со времени введения злокачественных клеток и тестируемых веществ (фигура 74).
Массу мыши измеряли с использованием весов и изменения в группе анализировали с использованием значений, измеренных дважды в неделю (фигура 75). Подтверждали, что не было значимых различий изменений массы тела в течение 3 недель между группой, в которой не инъецировали митохондрии, группой, в которой вводили митохондрии в отдельности, и группой, в которой инъецировали модифицированные митохондрии. Размер опухоли вычисляли, измеряя размер по длинной оси (длину) и короткой оси (ширину) опухоли с использованием калипера, а затем используя их в указанном выше математическом уравнении 1. Изменения в группе анализировали с использованием значений, измеренных дважды в неделю (фигура 76). Обнаружено, что размер опухоли значимо повышался с течением времени в группе, в которой не вводили митохондрии, в то время как в случае мышей, которым вводили митохондрии, повышение размера опухоли замедлялось с течением времени. Кроме того, подтверждали, что повышение размера опухоли значимо снижалось в группе, которой вводили митохондрии с нагруженным белком p53, по сравнению с группой, которой вводили митохондрии в отдельности (фигура 76).
Пример 22. Подтверждение эффекта модифицированных митохондрий в отношении ингибирования пролиферации злокачественных клеток кожи
Митохондрии, полученные, как описано выше, с которыми связан p53, вводили в клетки A431, являющиеся злокачественными клетками кожи, способом центрифугирования, а затем наблюдали пролиферацию клеток A431. В этом случае в качестве контрольной группы использовали физиологический раствор и в качестве контрольной тестовой группы использовали эквивалентное количество митохондрий, с которыми белок p53 не слит. Подтверждали, что митохондрии с нагруженным белком p53, белком, индуцирующим апоптоз, могут значительно ингибировать пролиферацию клеток A431 по сравнению с контрольной группой и группой, в которой использовали только митохондрии (фигура 76).
V. Подтверждение активности выделенных митохондрий
Пример 23. Подтверждение функции выделенных митохондрий: содержание АТФ
Для выделения внутриклеточных митохондрий из мезенхимальных стволовых клеток, полученных из пуповины (UC-MSC), осуществляли гомогенизацию с использованием шприца для разрушения клеток, а затем осуществляли непрерывное центрифугирование для получения митохондрий. Для подтверждения функции выделенных митохондрий концентрацию митохондриальных белков в выделенных митохондриях количественно анализировали посредством анализа BCA, получая 5 мкг митохондрий. Количество АТФ в митохондриях подтверждали с использованием набора для люминесцентного анализа CellTiter-Glo (Promega, Madison, WI).
Подготовленные митохондрии смешивали в 100 мкл PBS, а затем подготавливали в 96-луночном планшете и сравнивали с 100 мкл PBS, несодержащего митохондрии, в качестве контрольной группы. Тем же образом добавляли 100 мкл тестового раствора, включенного в набор, проводили реакцию и тщательно смешивали в течение 2 минут с помощью мешалки, а затем проводили реакцию при температуре окружающей среды в течение 10 минут, а затем измеряли количество АТФ с использованием люминесцентного спектрофотометра для чтения микропланшетов. Подтверждали, что АТФ повышался при включении митохондрий по сравнению с контрольной группой и подтверждали функцию митохондрий (фигура 78).
Пример 24. Подтверждение функции выделенных митохондрий: мембранный потенциал
Для подтверждения мембранного потенциала выделенных митохондрий использовали краситель JC-1 (Molecular Probes, кат. №1743159). Полученные митохондрии смешивали в 50 мкл PBS, а затем подготавливали в 96-луночном планшете. Получали группу PBS (50 мкл), не содержащего митохондрии, в качестве контрольной группы и группу обработки CCCP (R&D systems, CAS 555-60-2). CCCP, ионофор митохондрий, ингибирует функцию митохондрий посредством деполяризации митохондриального мембранного потенциала. Группа CCCP реагировала с выделенными митохондриями в количестве 50 мкМ в течение 10 минут при комнатной температуре.
Затем тем же образом проводили реакцию с красителем JC-1 (2 мкМ), а затем измеряли оптическую плотность, используя свойство, состоящее в том, что он имеет разный спектр в соответствии с концентрацией, генерируемый при изменении мембранного потенциала. При низких концентрациях он существует в виде мономера и демонстрирует зеленую флуоресценцию и при высоких концентрациях краситель агрегирует (J-агрегат), демонстрируя красную флуоресценцию. Митохондриальный мембранный потенциал анализировали, вычисляя соотношение зеленой флуоресценции и красной флуоресценции. После завершения реакции мембранный потенциал митохондрий измеряли с использованием флуоресцентного спектрофотометра для чтения микропланшетов (мономер: возбуждение 485/испускание 530, J-агрегат: возбуждение 535/испускание 590). Результаты показаны на фигуре 79.
Пример 25. Подтверждение степени повреждения выделенных митохондрий посредством подтверждения продукции mROS
Для подтверждения того, повреждены ли 5 мкг митохондрий, полученных, как описано выше, использовали красный краситель-индикатор MitoSOX (Invitrogen, кат. № M36008), с помощью которого можно анализировать митохондриальные активные формы кислорода в выделенных митохондриях. Полученные митохондрии смешивали в 50 мкл PBS, а затем подготавливали в 96-луночном планшете и сравнивали с 50 мкл PBS, несодержащего митохондрии, в качестве контрольной группы. Красный краситель MitoSOX смешивали в 50 мкл PBS до концентрации 10 мкМ и помещали в 96-луночный планшет (конечная концентрация 5 мкМ), а затем проводили реакцию в инкубаторе при 37°C, CO2 в течение 20 минут. После завершения реакции измеряли количество ROS в митохондриях с использованием спектрофотометра для чтения микропланшетов (возбуждение 510/испускание 580). Результаты показаны на фигуре 80.
VI. Подтверждение диссоциации желаемого белка, связанного с белком митохондриальной внешней мембраны, вне и внутри клеток
Пример 26. Подтверждение диссоциации желаемого белка, связанного с белком митохондриальной внешней мембраны, вне клетки
Для получения желаемого белка в свободной форме, когда активный белок, связанный с митохондриями, инъецировали в клетку, из E. coli получали слитый белок (TOM70-UB-p53 или TOM-UB-GFP) в форме, в которой белок убиквитин встроен между белком митохондриальной внешней мембраны и желаемым белком. Для подтверждения того, расщепляли ли последовательность убиквитина UBP1, расщепляющим убиквитин ферментом, проводили реакцию рекомбинантного слитого белка TOM70-UB-p53 с ферментом UBP1 при 37°C в течение 1 часа.
Затем в результате анализа посредством электрофореза в ПААГ с SDS подтверждали, что диссоциация белка убиквитина из слитого белка не происходила под действием UBP1. Это считали явлением интерференции структуры белка митохондриальной внешней мембраны и, таким образом, линкерный белок, состоящий из аминокислот глицина и серина, встраивали между белком митохондриальной внешней мембраны и белком убиквитином и получали новый слитый белок (TOM70-(GGGGS)3-UB-p53 или TOM70-(GGGGS)3-UB-GFP) посредством очистки из E. coli, а затем проводили реакцию с ферментом UBP1 при 37°C в течение 1 часа, как описано выше. В результате посредством электрофореза в ПААГ с SDS подтверждали, что 3'-конец убиквитина расщепляли ферментом UBP1, и диссоциировал только белок p53, как и ожидали (фигура 82).
Пример 26. Подтверждение диссоциация желаемого белка, связанного с белком митохондриальной внешней мембраны, внутри клеток
Если слитый белок (TOM70-(GGGGS)3-UB-p53 или TOM70-(GGGGS)3-UB-GFP), полученный в описанном выше примере, проникает в клетку в состоянии, связанном с митохондриями, определяли, диссоциировал ли активный белок под действием расщепляющего убиквитин фермента, присутствующего в клетке. Сначала проводили реакцию митохондрий, полученных из мезенхимальных клеток пуповинной крови, и слитого белка TOM70-(GGGGS)3-UB-GFP в течение 1 часа в микропробирке, чтобы позволить им связаться, а затем несвязанный слитый белок удаляли посредством центрифугирования, а затем дважды промывали буферным раствором PBS. В этом случае слитый белок (TOM70-(GGGGS)3-GFP), из которого удаляли убиквитин, использовали в качестве контрольной группы.
Затем белок, связанный с митохондриями, инъецировали в клетки MDA-MB-231, линию злокачественных клеток молочной железы, способом центрифугирования. Через день клетки MDA-MB-231 разрушали и фракционировали на митохондриальную часть и цитозольную часть, соответственно, с использованием дифференциального центрифугирования. В результате анализа посредством электрофореза в ПААГ с SDS и анализа вестерн-блоттинга обнаружено, что в случае слитого белка, в который включали убиквитин, белки GFP, диссоциировавшие от белка митохондриальной внешней мембраны, линкерный белок и убиквитин, главным образом, определяли в цитозольной части, и обнаружено, что в случае слитого белка, из которого удаляли убиквитин, белки GFP в форме, с которой связаны белок митохондриальной внешней мембраны и линкерный белок, главным образом, определяли в митохондриальной части (фигура 83).
В результате обнаружено, что, когда белок митохондриальной внешней мембраны-линкер-убиквитин-активный белок, связанный с митохондриями, инъецировали в клетки, участок соединения убиквитина и активного белка расщеплялся, и диссоциировавший активный белок высвобождался в цитоплазму, и обнаружено, что, таким образом, митохондрии можно использовать в виде средства доставки в качестве одного из способов эффективной доставки полезного белка в клетки.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Paean Biotechnology Inc.
<120> МОДИФИЦИРОВАННЫЕ МИТОХОНДРИИ И ИХ ПРИМЕНЕНИЕ
<130> PCB903024PBT/PCT
<150> KR 10-2018-0048486
<151> 2018-04-26
<160> 92
<170> KoPatentIn 3.0
<210> 1
<211> 39
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер T2p53
<400> 1
aaaaaaccgc ggtggtgagg agccgcagtc agatcctag
39
<210> 2
<211> 35
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер Xp53
<400> 2
aaaaaactcg agtgagtctg agtcaggccc ttctg
35
<210> 3
<211> 1186
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность, кодирующая p53
<400> 3
ccgcggtggt gaggagccgc agtcagatcc tagcgtcgag ccccctctga gtcaggaaac
60
attttcagac ctgtggaaac tacttcctga aaacaacgtt ctgtccccct tgccgtccca
120
agcaatggat gatttgatgc tgtccccgga cgatattgaa caatggttca ctgaagaccc
180
aggtccagat gaagctccca gaatgccaga ggctgctccc cgcgtggccc ctgcaccagc
240
agctcctaca ccggcggccc ctgcaccagc cccctcctgg cccctgtcat cttctgtccc
300
ttcccagaaa acctaccagg gcagctacgg tttccgtctg ggcttcttgc attctgggac
360
agccaagtct gtgacttgca cgtactcccc tgccctcaac aagatgtttt gccaactggc
420
caagacctgc cctgtgcagc tgtgggttga ttccacaccc ccgcccggca cccgcgtccg
480
cgccatggcc atctacaagc agtcacagca catgacggag gttgtgaggc gctgccccca
540
ccatgagcgc tgctcagata gcgatggtct ggcccctcct cagcatctta tccgagtgga
600
aggaaatttg cgtgtggagt atttggatga cagaaacact tttcgacata gtgtggtggt
660
gccctatgag ccgcctgagg ttggctctga ctgtaccacc atccactaca actacatgtg
720
taacagttcc tgcatgggcg gcatgaaccg gaggcccatc ctcaccatca tcacactgga
780
agactccagt ggtaatctac tgggacggaa cagctttgag gtgcgtgttt gtgcctgtcc
840
tgggagagac cggcgcacag aggaagagaa tctccgcaag aaaggggagc ctcaccacga
900
gctgccccca gggagcacta agcgagcact gcccaacaac accagctcct ctccccagcc
960
aaagaagaaa ccactggatg gagaatattt cacccttcag atccgtgggc gtgagcgctt
1020
cgagatgttc cgagagctga atgaggcctt ggaactcaag gatgcccagg ctgggaagga
1080
gccagggggg agcagggctc actccagcca cctgaagtcc aaaaagggtc agtctacctc
1140
ccgccataaa aaactcatgt tcaagacaga agggcctgac tcagac
1186
<210> 4
<211> 35
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер NdeUB
<400> 4
ggattccata tgcaactttt cgtcaaaact ctaac
35
<210> 5
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер T2UB
<400> 5
atgaccaccg cggagtctca acaccaa
27
<210> 6
<211> 1460
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность, кодирующая UB-p53
<400> 6
atgggcagca gccatcatca tcatcatcac agcagcggcc tggtgccgcg cggcagccat
60
atgcaaatct tcgtcaaaac tctaacaggg aagactataa ccctagaggt tgaaccatcc
120
gacactattg aaaacgtcaa agctaaaatt caagataaag aaggtatccc tccggatcag
180
cagagattga tttttgctgg taagcaacta gaagatggta gaaccttgtc tgactacaac
240
atccaaaagg aatctactct tcacttggtg ttgagactcc gcggtggtga ggagccgcag
300
tcagatccta gcgtcgagcc ccctctgagt caggaaacat tttcagacct gtggaaacta
360
cttcctgaaa acaacgttct gtcccccttg ccgtcccaag caatggatga tttgatgctg
420
tccccggacg atattgaaca atggttcact gaagacccag gtccagatga agctcccaga
480
atgccagagg ctgctccccg cgtggcccct gcaccagcag ctcctacacc ggcggcccct
540
gcaccagccc cctcctggcc cctgtcatct tctgtccctt cccagaaaac ctaccagggc
600
agctacggtt tccgtctggg cttcttgcat tctgggacag ccaagtctgt gacttgcacg
660
tactcccctg ccctcaacaa gatgttttgc caactggcca agacctgccc tgtgcagctg
720
tgggttgatt ccacaccccc gcccggcacc cgcgtccgcg ccatggccat ctacaagcag
780
tcacagcaca tgacggaggt tgtgaggcgc tgcccccacc atgagcgctg ctcagatagc
840
gatggtctgg cccctcctca gcatcttatc cgagtggaag gaaatttgcg tgtggagtat
900
ttggatgaca gaaacacttt tcgacatagt gtggtggtgc cctatgagcc gcctgaggtt
960
ggctctgact gtaccaccat ccactacaac tacatgtgta acagttcctg catgggcggc
1020
atgaaccgga ggcccatcct caccatcatc acactggaag actccagtgg taatctactg
1080
ggacggaaca gctttgaggt gcgtgtttgt gcctgtcctg ggagagaccg gcgcacagag
1140
gaagagaatc tccgcaagaa aggggagcct caccacgagc tgcccccagg gagcactaag
1200
cgagcactgc ccaacaacac cagctcctct ccccagccaa agaagaaacc actggatgga
1260
gaatatttca cccttcagat ccgtgggcgt gagcgcttcg agatgttccg agagctgaat
1320
gaggccttgg aactcaagga tgcccaggct gggaaggagc caggggggag cagggctcac
1380
tccagccacc tgaagtccaa aaagggtcag tctacctccc gccataaaaa actcatgttc
1440
aagacagaag ggcctgatag
1460
<210> 7
<211> 59
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер NdeTOM70
<400> 7
gaattccata tgaaaagttt tataactcgg aataaaactg caattttcgc aactgttgc
59
<210> 8
<211> 38
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер TOM70-AS
<400> 8
ggtgcatact actattatca aacttttcgt caaaactc
38
<210> 9
<211> 36
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер TOM70UB-S
<400> 9
ggctacgtat ttatttccaa cttttcgtca aaactc
36
<210> 10
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер T2UB-AS
<400> 10
ggcaccaccg cggagtctca acac
24
<210> 11
<211> 1515
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность, кодирующая TOM70-UB-p53
<400> 11
atgaaaagtt ttataactcg gaataaaact gcaattttcg caactgttgc tgctacggga
60
accgctattg gtgcatacta ctattatcaa atcttcgtca aaactctaac agggaagact
120
ataaccctag aggttgaacc atccgacact attgaaaacg tcaaagctaa aattcaagat
180
aaagaaggta tccctccgga tcagcagaga ttgatttttg ctggtaagca actagaagat
240
ggtagaacct tgtctgacta caacatccaa aaggaatcta ctcttcactt ggtgttgaga
300
ctccgcggtg gtgaggagcc gcagtcagat cctagcgtcg agccccctct gagtcaggaa
360
acattttcag acctgtggaa actacttcct gaaaacaacg ttctgtcccc cttgccgtcc
420
caagcaatgg atgatttgat gctgtccccg gacgatattg aacaatggtt cactgaagac
480
ccaggtccag atgaagctcc cagaatgcca gaggctgctc cccgcgtggc ccctgcacca
540
gcagctccta caccggcggc ccctgcacca gccccctcct ggcccctgtc atcttctgtc
600
ccttcccaga aaacctacca gggcagctac ggtttccgtc tgggcttctt gcattctggg
660
acagccaagt ctgtgacttg cacgtactcc cctgccctca acaagatgtt ttgccaactg
720
gccaagacct gccctgtgca gctgtgggtt gattccacac ccccgcccgg cacccgcgtc
780
cgcgccatgg ccatctacaa gcagtcacag cacatgacgg aggttgtgag gcgctgcccc
840
caccatgagc gctgctcaga tagcgatggt ctggcccctc ctcagcatct tatccgagtg
900
gaaggaaatt tgcgtgtgga gtatttggat gacagaaaca cttttcgaca tagtgtggtg
960
gtgccctatg agccgcctga ggttggctct gactgtacca ccatccacta caactacatg
1020
tgtaacagtt cctgcatggg cggcatgaac cggaggccca tcctcaccat catcacactg
1080
gaagactcca gtggtaatct actgggacgg aacagctttg aggtgcgtgt ttgtgcctgt
1140
cctgggagag accggcgcac agaggaagag aatctccgca agaaagggga gcctcaccac
1200
gagctgcccc cagggagcac taagcgagca ctgcccaaca acaccagctc ctctccccag
1260
ccaaagaaga aaccactgga tggagaatat ttcacccttc agatccgtgg gcgtgagcgc
1320
ttcgagatgt tccgagagct gaatgaggcc ttggaactca aggatgccca ggctgggaag
1380
gagccagggg ggagcagggc tcactccagc cacctgaagt ccaaaaaggg tcagtctacc
1440
tcccgccata aaaaactcat gttcaagaca gaagggcctg actcagacct cgagcaccac
1500
caccaccacc actag
1515
<210> 12
<211> 59
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер TOM70(G)3-AS
<400> 12
gcccccggat cctccacccc cgcttccgcc acctccataa tagtagtatg caccaatag
59
<210> 13
<211> 32
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер (G)3UB-S
<400> 13
ggtggaggat ccgggggcgc ggaagccaaa tc
32
<210> 14
<211> 32
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер Xp53(noT)
<400> 14
aaaaaactcg aggtctgagt caggcccttc tg
32
<210> 15
<211> 1560
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность, кодирующая
TOM70-(GGGGS)3-UB-p53
<400> 15
atgaaaagtt ttataactcg gaataaaact gcaattttcg caactgttgc tgctacggga
60
accgctattg gtgcatacta ctattatgga ggtggcggaa gcgggggtgg aggatccggg
120
ggcggcggaa gccaaatctt cgtcaaaact ctaacaggga agactataac cctagaggtt
180
gaaccatccg acactattga aaacgtcaaa gctaaaattc aagataaaga aggtatccct
240
ccggatcagc agagattgat ttttgctggt aagcaactag aagatggtag aaccttgtct
300
gactacaaca tccaaaagga atctactctt cacttggtgt tgagactccg cggtggtgag
360
gagccgcagt cagatcctag cgtcgagccc cctctgagtc aggaaacatt ttcagacctg
420
tggaaactac ttcctgaaaa caacgttctg tcccccttgc cgtcccaagc aatggatgat
480
ttgatgctgt ccccggacga tattgaacaa tggttcactg aagacccagg tccagatgaa
540
gctcccagaa tgccagaggc tgctccccgc gtggcccctg caccagcagc tcctacaccg
600
gcggcccctg caccagcccc ctcctggccc ctgtcatctt ctgtcccttc ccagaaaacc
660
taccagggca gctacggttt ccgtctgggc ttcttgcatt ctgggacagc caagtctgtg
720
acttgcacgt actcccctgc cctcaacaag atgttttgcc aactggccaa gacctgccct
780
gtgcagctgt gggttgattc cacacccccg cccggcaccc gcgtccgcgc catggccatc
840
tacaagcagt cacagcacat gacggaggtt gtgaggcgct gcccccacca tgagcgctgc
900
tcagatagcg atggtctggc ccctcctcag catcttatcc gagtggaagg aaatttgcgt
960
gtggagtatt tggatgacag aaacactttt cgacatagtg tggtggtgcc ctatgagccg
1020
cctgaggttg gctctgactg taccaccatc cactacaact acatgtgtaa cagttcctgc
1080
atgggcggca tgaaccggag gcccatcctc accatcatca cactggaaga ctccagtggt
1140
aatctactgg gacggaacag ctttgaggtg cgtgtttgtg cctgtcctgg gagagaccgg
1200
cgcacagagg aagagaatct ccgcaagaaa ggggagcctc accacgagct gcccccaggg
1260
agcactaagc gagcactgcc caacaacacc agctcctctc cccagccaaa gaagaaacca
1320
ctggatggag aatatttcac ccttcagatc cgtgggcgtg agcgcttcga gatgttccga
1380
gagctgaatg aggccttgga actcaaggat gcccaggctg ggaaggagcc aggggggagc
1440
agggctcact ccagccacct gaagtccaaa aagggtcagt ctacctcccg ccataaaaaa
1500
ctcatgttca agacagaagg gcctgactca gacctcgagc accaccacca ccaccactag
1560
1560
<210> 16
<211> 51
<212> ДНК
<213> Искусственная последовательность
<220>
<223> B(G)3p53
<400> 16
ggtggaggat ccgggggcgg cggaagcgag gagccgcagt cagatcctag c
51
<210> 17
<211> 1335
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность, кодирующая
TOM70-(GGGGS)3-p53
<400> 17
atgaaaagtt ttataactcg gaataaaact gcaattttcg caactgttgc tgctacggga
60
accgctattg gtgcatacta ctattatgga ggtggcggaa gcgggggtgg aggatccggg
120
ggcggcggaa gcgaggagcc gcagtcagat cctagcgtcg agccccctct gagtcaggaa
180
acattttcag acctgtggaa actacttcct gaaaacaacg ttctgtcccc cttgccgtcc
240
caagcaatgg atgatttgat gctgtccccg gacgatattg aacaatggtt cactgaagac
300
ccaggtccag atgaagctcc cagaatgcca gaggctgctc cccgcgtggc ccctgcacca
360
gcagctccta caccggcggc ccctgcacca gccccctcct ggcccctgtc atcttctgtc
420
ccttcccaga aaacctacca gggcagctac ggtttccgtc tgggcttctt gcattctggg
480
acagccaagt ctgtgacttg cacgtactcc cctgccctca acaagatgtt ttgccaactg
540
gccaagacct gccctgtgca gctgtgggtt gattccacac ccccgcccgg cacccgcgtc
600
cgcgccatgg ccatctacaa gcagtcacag cacatgacgg aggttgtgag gcgctgcccc
660
caccatgagc gctgctcaga tagcgatggt ctggcccctc ctcagcatct tatccgagtg
720
gaaggaaatt tgcgtgtgga gtatttggat gacagaaaca cttttcgaca tagtgtggtg
780
gtgccctatg agccgcctga ggttggctct gactgtacca ccatccacta caactacatg
840
tgtaacagtt cctgcatggg cggcatgaac cggaggccca tcctcaccat catcacactg
900
gaagactcca gtggtaatct actgggacgg aacagctttg aggtgcgtgt ttgtgcctgt
960
cctgggagag accggcgcac agaggaagag aatctccgca agaaagggga gcctcaccac
1020
gagctgcccc cagggagcac taagcgagca ctgcccaaca acaccagctc ctctccccag
1080
ccaaagaaga aaccactgga tggagaatat ttcacccttc agatccgtgg gcgtgagcgc
1140
ttcgagatgt tccgagagct gaatgaggcc ttggaactca aggatgccca ggctgggaag
1200
gagccagggg ggagcagggc tcactccagc cacctgaagt ccaaaaaggg tcagtctacc
1260
tcccgccata aaaaactcat gttcaagaca gaagggcctg actcagacct cgagcaccac
1320
caccaccacc actag
1335
<210> 18
<211> 32
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер Xp53(noT)
<400> 18
aaaaaactcg aggtctgagt caggcccttc tg
32
<210> 19
<211> 36
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер XTOM7
<400> 19
aaaaaactcg agtttgccat tcgctggggc tttatc
36
<210> 20
<211> 38
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер LTOM7
<400> 20
aaaaaagtcg acttatcccc aaagtaggct caaaacag
38
<210> 21
<211> 1578
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность, кодирующая UB-p53-TOM7
<400> 21
atgggcagca gccatcatca tcatcatcac agcagcggcc tggtgccgcg cggcagccat
60
atgcaaatct tcgtcaaaac tctaacaggg aagactataa ccctagaggt tgaaccatcc
120
gacactattg aaaacgtcaa agctaaaatt caagataaag aaggtatccc tccggatcag
180
cagagattga tttttgctgg taagcaacta gaagatggta gaaccttgtc tgactacaac
240
atccaaaagg aatctactct tcacttggtg ttgagactcc gcggtggtga ggagccgcag
300
tcagatccta gcgtcgagcc ccctctgagt caggaaacat tttcagacct atggaaacta
360
cttcctgaaa acaacgttct gtcccccttg ccgtcccaag caatggatga tttgatgctg
420
tccccggacg atattgaaca atggttcact gaagacccag gtccagatga agctcccaga
480
atgccagagg ctgctccccg cgtggcccct gcaccagcag ctcctacacc ggcggcccct
540
gcaccagccc cctcctggcc cctgtcatct tctgtccctt cccagaaaac ctaccagggc
600
agctacggtt tccgtctggg cttcttgcat tctgggacag ccaagtctgt gacttgcacg
660
tactcccctg ccctcaacaa gatgttttgc caactggcca agacctgccc tgtgcagctg
720
tgggttgatt ccacaccccc gcccggcacc cgcgtccgcg ccatggccat ctacaagcag
780
tcacagcaca tgacggaggt tgtgaggcgc tgcccccacc atgagcgctg ctcagatagc
840
gatggtctgg cccctcctca gcatcttatc cgagtggaag gaaatttgcg tgtggagtat
900
ttggatgaca gaaacacttt tcgacatagt gtggtggtgc cctatgagcc gcctgaggtt
960
ggctctgact gtaccaccat ccactacaac tacatgtgta acagttcctg catgggcggc
1020
atgaaccgga ggcccatcct caccatcatc acactggaag actccagtgg taatctactg
1080
ggacggaaca gctttgaggt gcatgtttgt gcctgtcctg ggagagaccg gcgcacagag
1140
gaagagaatc tccgcaagaa aggggagcct caccacgagc tgcccccagg gagcactaag
1200
cgagcactgt ccaacaacac cagctcctct ccccagccaa agaagaaacc actggatgga
1260
gaatatttca cccttcagat ccgtgggcgt gagcgcttcg agatgttccg agagctgaat
1320
gaggccttgg aactcaagga tgcccaggct gggaaggagc caggggggag cagggctcac
1380
tccagccacc tgaagtccaa aaagggtcag tctacctccc gccataaaaa actcatgttc
1440
aagacagaag ggcctgactc agacctcgag tttgccattc gctggggctt tatccctctt
1500
gtgatttacc tgggatttaa gaggggtgca gatcccggaa tgcctgaacc aactgttttg
1560
agcctacttt ggggatag
1578
<210> 22
<211> 35
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер Rp53
<400> 22
aaaaaagaat tcatggtctg agtcaggccc ttctg
35
<210> 23
<211> 1266
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность, кодирующая p53-myc/His
<400> 23
atggaggagc cgcagtcaga tcctagcgtc gagccccctc tgagtcagga aacattttca
60
gacctgtgga aactacttcc tgaaaacaac gttctgtccc ccttgccgtc ccaagcaatg
120
gatgatttga tgctgtcccc ggacgatatt gaacaatggt tcactgaaga cccaggtcca
180
gatgaagctc ccagaatgcc agaggctgct ccccgcgtgg cccctgcacc agcagctcct
240
acaccggcgg cccctgcacc agccccctcc tggcccctgt catcttctgt cccttcccag
300
aaaacctacc agggcagcta cggtttccgt ctgggcttct tgcattctgg gacagccaag
360
tctgtgactt gcacgtactc ccctgccctc aacaagatgt tttgccaact ggccaagacc
420
tgccctgtgc agctgtgggt tgattccaca cccccgcccg gcacccgcgt ccgcgccatg
480
gccatctaca agcagtcaca gcacatgacg gaggttgtga ggcgctgccc ccaccatgag
540
cgctgctcag atagcgatgg tctggcccct cctcagcatc ttatccgagt ggaaggaaat
600
ttgcgtgtgg agtatttgga tgacagaaac acttttcgac atagtgtggt ggtgccctat
660
gagccgcctg aggttggctc tgactgtacc accatccact acaactacat gtgtaacagt
720
tcctgcatgg gcggcatgaa ccggaggccc atcctcacca tcatcacact ggaagactcc
780
agtggtaatc tactgggacg gaacagcttt gaggtgcgtg tttgtgcctg tcctgggaga
840
gaccggcgca cagaggaaga gaatctccgc aagaaagggg agcctcacca cgagctgccc
900
ccagggagca ctaagcgagc actgcccaac aacaccagct cctctcccca gccaaagaag
960
aaaccactgg atggagaata tttcaccctt cagatccgtg ggcgtgagcg cttcgagatg
1020
ttccgagagc tgaatgaggc cttggaactc aaggatgccc aggctgggaa ggagccaggg
1080
gggagcaggg ctcactccag ccacctgaag tccaaaaagg gtcagtctac ctcccgccat
1140
aaaaaactca tgttcaagac agaagggcct gactcagacc tcgagtctag agggcccttc
1200
gaacaaaaac tcatctcaga agaggatctg aatatgcata ccggtcatca tcaccatcac
1260
cattga
1266
<210> 24
<211> 51
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер T2GZMB
<400> 24
aaaaaaccgc ggtggtatca tcgggggaca tgaggcacat gaggccaagc c
51
<210> 25
<211> 38
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер XGZMB(noT)
<400> 25
aaaaaactcg aggtagcgtt tcatggtttt ctttatcc
38
<210> 26
<211> 691
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность, кодирующая GranzymeB
<400> 26
ccgcggtggt atcatcgggg gacatgaggc caagccccac tcccgcccct acatggctta
60
tcttatgatc tgggatcaga agtctctgaa gaggtgcggt ggcttcctga tacaagacga
120
cttcgtgctg acagctgctc actgttgggg aagctccata aatgtcacct tgggggccca
180
caatatcaaa gaacaggagc cgacccagca gtttatccct gtgaaaagac ccatccccca
240
tccagcctat aatcctaaga acttctccaa cgacatcatg ctactgcagc tggagagaaa
300
ggccaagcgg accagagctg tgcagcccct caggctacct agcaacaagg cccaggtgaa
360
gccagggcag acatgcagtg tggccggctg ggggcagacg gcccccctgg gaaaacactc
420
acacacacta caagaggtga agatgacagt gcaggaagat cgaaagtgcg aatctgactt
480
acgccattat tacgacagta ccattgagtt gtgcgtgggg gacccagaga ttaaaaagac
540
ttcctttaag ggggactctg gaggccctct tgtgtgtaac aaggtggccc agggcattgt
600
ctcctatgga cgaaacaatg gcatgcctcc acgagcctgc accaaagtct caagctttgt
660
acactggata aagaaaacca tgaaacgcta c
691
<210> 27
<211> 1065
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность, кодирующая
TOM70-(GGGGS)3-UB-гранзим B
<400> 27
atgaaaagtt ttataactcg gaataaaact gcaattttcg caactgttgc tgctacggga
60
accgctattg gtgcatacta ctattatgga ggtggcggaa gcgggggtgg aggatccggg
120
ggcggcggaa gccaaatctt cgtcaaaact ctaacaggga agactataac cctagaggtt
180
gaaccatccg acactattga aaacgtcaaa gctaaaattc aagataaaga aggtatccct
240
ccggatcagc agagattgat ttttgctggt aagcaactag aagatggtag aaccttgtct
300
gactacaaca tccaaaagga atctactctt cacttggtgt tgagactccg cggtggtatc
360
atcgggggac atgaggccaa gccccactcc cgcccctaca tggcttatct tatgatctgg
420
gatcagaagt ctctgaagag gtgcggtggc ttcctgatac gagacgactt cgtgctgaca
480
gctgctcact gttggggaag ctccataaat gtcaccttgg gggcccacaa tatcaaagaa
540
caggagccga cccagcagtt tatccctgtg aaaagaccca tcccccatcc agcctataat
600
cctaagaact tctccaacga catcatgcta ctgcagctgg agagaaaggc caagcggacc
660
agagctgtgc agcccctcag gctacctagc aacaaggccc aggtgaagcc agggcagaca
720
tgcagtgtgg ccggctgggg gcagacggcc cccctgggaa aacactcaca cacactacaa
780
gaggtgaaga tgacagtgca ggaagatcga aagtgcgaat ctgacttacg ccattattac
840
gacagtacca ttgagttgtg cgtgggggac ccagagatta aaaagacttc ctttaagggg
900
gactctggag gccctcttgt gtgtaacaag gtggcccagg gcattgtctc ctatggacga
960
aacaatggca tgcctccacg agcctgcacc aaagtctcaa gctttgtaca ctggataaag
1020
aaaaccatga aacgctacct cgagcaccac caccaccacc actag
1065
<210> 28
<211> 1083
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность, кодирующая UB-гранзим
B-TOM7
<400> 28
atgggcagca gccatcatca tcatcatcac agcagcggcc tggtgccgcg cggcagccat
60
atgcaaatct tcgtcaaaac tctaacaggg aagactataa ccctagaggt tgaaccatcc
120
gacactattg aaaacgtcaa agctaaaatt caagataaag aaggtatccc tccggatcag
180
cagagattga tttttgctgg taagcaacta gaagatggta gaaccttgtc tgactacaac
240
atccaaaagg aatctactct tcacttggtg ttgagactcc gcggtggtat catcggggga
300
catgaggcca agccccactc ccgcccctac atggcttatc ttatgatctg ggatcagaag
360
tctctgaaga ggtgcggtgg cttcctgata cgagacgact tcgtgctgac agctgctcac
420
tgttggggaa gctccataaa tgtcaccttg ggggcccaca atatcaaaga acaggagccg
480
acccagcagt ttatccctgt gaaaagaccc atcccccatc cagcctataa tcctaagaac
540
ttctccaacg acatcatgct actgcagctg gagagaaagg ccaagcggac cagagctgtg
600
cagcccctca ggctacctag caacaaggcc caggtgaagc cagggcagac atgcagtgtg
660
gccggctggg ggcagacggc ccccctggga aaacactcac acacactaca agaggtgaag
720
atgacagtgc aggaagatcg aaagtgcgaa tctgacttac gccattatta cgacagtacc
780
attgagttgt gcgtggggga cccagagatt aaaaagactt cctttaaggg ggactctgga
840
ggccctcttg tgtgtaacaa ggtggcccag ggcattgtct cctatggacg aaacaatggc
900
atgcctccac gagcctgcac caaagtctca agctttgtac actggataaa gaaaaccatg
960
aaacgctacc tcgagtttgc cattcgctgg ggctttatcc ctcttgtgat ttacctggga
1020
tttaagaggg gtgcagatcc cggaatgcct gaaccaactg ttttgagcct actttgggga
1080
tag
1083
<210> 29
<211> 39
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер T2RKIP
<400> 29
aaaaaaccgc ggtggtccgg tggacctcag caagtggtc
39
<210> 30
<211> 41
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер XRKIP(noT)
<400> 30
aaaaaactcg agcttcccag acagctgctc gtacagtttg g
41
<210> 31
<211> 568
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность, кодирующая RKIP
<400> 31
ccgcggtggt ccggtggacc tcagcaagtg gtccgggccc ttgagcctgc aagaagtgga
60
cgagcagccg cagcacccac tgcatgtcac ctacgccggg gcggcggtgg acgagctggg
120
caaagtgctg acgcccaccc aggttaagaa tagacccacc agcatttcgt gggatggtct
180
tgattcaggg aagctctaca ccttggtcct gacagacccg gatgctccca gcaggaagga
240
tcccaaatac agagaatggc atcatttcct ggtggtcaac atgaagggca atgacatcag
300
cagtggcaca gtcctctccg attatgtggg ctcggggcct cccaagggca caggcctcca
360
ccgctatgtc tggctggttt acgagcagga caggccgcta aagtgtgacg agcccatcct
420
cagcaaccga tctggagacc accgtggcaa attcaaggtg gcgtccttcc gtaaaaagta
480
tgagctcagg gccccggtgg ctggcacgtg ttaccaggcc gagtgggatg actatgtgcc
540
caaactgtac gagcagctgt ctgggaag
568
<210> 32
<211> 942
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность, кодирующая
TOM70-(GGGGS)3-UB-RKIP
<400> 32
atgaaaagtt ttataactcg gaataaaact gcaattttcg caactgttgc tgctacggga
60
accgctattg gtgcatacta ctattatgga ggtggcggaa gcgggggtgg aggatccggg
120
ggcggcggaa gccaaatctt cgtcaaaact ctaacaggga agactataac cctagaggtt
180
gaaccatccg acactattga aaacgtcaaa gctaaaattc aagataaaga aggtatccct
240
ccggatcagc agagattgat ttttgctggt aagcaactag aagatggtag aaccttgtct
300
gactacaaca tccaaaagga atctactctt cacttggtgt tgagactccg cggtggtccg
360
gtggacctca gcaagtggtc cgggcccttg agcctgcaag aagtggacga gcagccgcag
420
cacccactgc atgtcaccta cgccggggcg gcggtggacg agctgggcaa agtgctgacg
480
cccacccagg ttaagaatag acccaccagc atttcgtggg atggtcttga ttcagggaag
540
ctctacacct tggtcctgac agacccggat gctcccagca ggaaggatcc caaatacaga
600
gaatggcatc atttcctggt ggtcaacatg aagggcaatg acatcagcag tggcacagtc
660
ctctccgatt atgtgggctc ggggcctccc aagggcacag gcctccaccg ctatgtctgg
720
ctggtttacg agcaggacag gccgctaaag tgtgacgagc ccatcctcag caaccgatct
780
ggagaccacc gtggcaaatt caaggtggcg tccttccgta aaaagtatga gctcagggcc
840
ccggtggctg gcacgtgtta ccaggccgag tgggatgact atgtgcccaa actgtacgag
900
cagctgtctg ggaagctcga gcaccaccac caccaccact ag
942
<210> 33
<211> 42
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер T2PTEN
<400> 33
aaaaaaccgc ggtggtacag ccatcatcaa agagatcgtt ag
42
<210> 34
<211> 36
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер XPTEN(noT)
<400> 34
aaaaaactcg aggacttttg taatttgtgt atgctg
36
<210> 35
<211> 1216
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность, кодирующая PTEN
<400> 35
ccgcggtggt acagccatca tcaaagagat cgttagcaga aacaaaagga gatatcaaga
60
ggatggattc gacttagact tgacctatat ttatccaaac attattgcta tgggatttcc
120
tgcagaaaga cttgaaggcg tatacaggaa caatattgat gatgtagtaa ggtttttgga
180
ttcaaagcat aaaaaccatt acaagatata caatctttgt gctgaaagac attatgacac
240
cgccaaattt aattgcagag ttgcacaata tccttttgaa gaccataacc caccacagct
300
agaacttatc aaaccctttt gtgaagatct tgaccaatgg ctaagtgaag atgacaatca
360
tgttgcagca attcactgta aagctggaaa gggacgaact ggtgtaatga tatgtgcata
420
tttattacat cggggcaaat ttttaaaggc acaagaggcc ctagatttct atggggaagt
480
aaggaccaga gacaaaaagg gagtaactat tcccagtcag aggcgctatg tgtattatta
540
tagctacctg ttaaagaatc atctggatta tagaccagtg gcactgttgt ttcacaagat
600
gatgtttgaa actattccaa tgttcagtgg cggaacttgc aatcctcagt ttgtggtctg
660
ccagctaaag gtgaagatat attcctccaa ttcaggaccc acacgacggg aagacaagtt
720
catgtacttt gagttccctc agccgttacc tgtgtgtggt gatatcaaag tagagttctt
780
ccacaaacag aacaagatgc taaaaaagga caaaatgttt cacttttggg taaatacatt
840
cttcatacca ggaccagagg aaacctcaga aaaagtagaa aatggaagtc tatgtgatca
900
agaaatcgat agcatttgca gtatagagcg tgcagataat gacaaggaat atctagtact
960
tactttaaca aaaaatgatc ttgacaaagc aaataaagac aaagccaacc gatacttttc
1020
tccaaatttt aaggtgaagc tgtacttcac aaaaacagta gaggagccgt caaatccaga
1080
ggctagcagt tcaacttctg taacaccaga tgttagtgac aatgaacctg atcattatag
1140
atattctgac accactgact ctgatccaga gaatgaacct tttgatgaag atcagcatac
1200
acaaattaca aaagtc
1216
<210> 36
<211> 1590
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность, кодирующая
TOM70-(GGGGS)3-UB-PTEN
<400> 36
atgaaaagtt ttataactcg gaataaaact gcaattttcg caactgttgc tgctacggga
60
accgctattg gtgcatacta ctattatgga ggtggcggaa gcgggggtgg aggatccggg
120
ggcggcggaa gccaaatctt cgtcaaaact ctaacaggga agactataac cctagaggtt
180
gaaccatccg acactattga aaacgtcaaa gctaaaattc aagataaaga aggtatccct
240
ccggatcagc agagattgat ttttgctggt aagcaactag aagatggtag aaccttgtct
300
gactacaaca tccaaaagga atctactctt cacttggtgt tgagactccg cggtggtaca
360
gccatcatca aagagatcgt tagcagaaac aaaaggagat atcaagagga tggattcgac
420
ttagacttga cctatattta tccaaacatt attgctatgg gatttcctgc agaaagactt
480
gaaggcgtat acaggaacaa tattgatgat gtagtaaggt ttttggattc aaagcataaa
540
aaccattaca agatatacaa tctttgtgct gaaagacatt atgacaccgc caaatttaat
600
tgcagagttg cacaatatcc ttttgaagac cataacccac cacagctaga acttatcaaa
660
cccttttgtg aagatcttga ccaatggcta agtgaagatg acaatcatgt tgcagcaatt
720
cactgtaaag ctggaaaggg acgaactggt gtaatgatat gtgcatattt attacatcgg
780
ggcaaatttt taaaggcaca agaggcccta gatttctatg gggaagtaag gaccagagac
840
aaaaagggag taactattcc cagtcagagg cgctatgtgt attattatag ctacctgtta
900
aagaatcatc tggattatag accagtggca ctgttgtttc acaagatgat gtttgaaact
960
attccaatgt tcagtggcgg aacttgcaat cctcagtttg tggtctgcca gctaaaggtg
1020
aagatatatt cctccaattc aggacccaca cgacgggaag acaagttcat gtactttgag
1080
ttccctcagc cgttacctgt gtgtggtgat atcaaagtag agttcttcca caaacagaac
1140
aagatgctaa aaaaggacaa aatgtttcac ttttgggtaa atacattctt cataccagga
1200
ccagaggaaa cctcagaaaa agtagaaaat ggaagtctat gtgatcaaga aatcgatagc
1260
atttgcagta tagagcgtgc agataatgac aaggaatatc tagtacttac tttaacaaaa
1320
aatgatcttg acaaagcaaa taaagacaaa gccaaccgat acttttctcc aaattttaag
1380
gtgaagctgt acttcacaaa aacagtagag gagccgtcaa atccagaggc tagcagttca
1440
acttctgtaa caccagatgt tagtgacaat gaacctgatc attatagata ttctgacacc
1500
actgactctg atccagagaa tgaacctttt gatgaagatc agcatacaca aattacaaaa
1560
gtcctcgagc accaccacca ccaccactag
1590
<210> 37
<211> 739
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность, кодирующая scFvHER2
<400> 37
ccgcggtggt gaagtgcaac ttgttgagag tggcggagga ctggtccaac cgggcggttc
60
acttaggctc tcatgtgcag cttcagggtt cacgttcacg gactatacaa tggactgggt
120
gaggcaagcc cctggtaagg gactggagtg ggttgctgac gttaacccta attccggtgg
180
gtccatctac aaccagcgat tcaagggacg atttactctt tcagtcgaca gaagcaaaaa
240
caccctctac ctccagatga actccttgcg ggcagaggat acagcggtct actattgcgc
300
gagaaacttg ggaccaagct tctacttcga ctactggggg caaggaacgc ttgttacggt
360
ttcaagcgga ggtggaggaa gtggaggtgg cggttccggc ggtggcggtt cagatataca
420
gatgacccaa tcacccagtt ctcttagcgc gtctgtaggc gacagggtaa ccataacctg
480
caaggcgtcc caggacgtgt caattggagt tgcctggtat cagcaaaaac ctgggaaagc
540
tccgaagctc ctgatttaca gcgcatctta ccgatatact ggtgtccctt caaggttcag
600
tggcagtgga tctgggacag actttacgct tactatcagc agtctgcaac ctgaggattt
660
cgcgacctac tactgtcagc agtattacat ctatccgtac acgttcggtc aaggtacaaa
720
ggtagaaata aaacgcact
739
<210> 38
<211> 1125
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность, кодирующая UB-scFvHER2-TOM7
<400> 38
atgggcagca gccatcatca tcatcatcac agcagcggcc tggtgccgcg cggcagccat
60
atgcaaatct tcgtcaaaac tctaacaggg aagactataa ccctagaggt tgaaccatcc
120
gacactattg aaaacgtcaa agctaaaatt caagataaag aaggtatccc tccggatcag
180
cagagattga tttttgctgg taagcaacta gaagatggta gaaccttgtc tgactacaac
240
atccaaaagg aatctactct tcacttggtg ttgagactcc gcggtggtga agtgcaactt
300
gttgagagtg gcggaggact ggtccaaccg ggcggttcac ttaggctctc atgtgcagct
360
tcagggttca cgttcacgga ctatacaatg gactgggtga ggcaagcccc tggtaaggga
420
ctggagtggg ttgctgacgt taaccctaat tccggtgggt ccatctacaa ccagcgattc
480
aagggacgat ttactctttc agtcgacaga agcaaaaaca ccctctacct ccagatgaac
540
tccttgcggg cagaggatac agcggtctac tattgcgcga gaaacttggg accaagcttc
600
tacttcgact actgggggca aggaacgctt gttacggttt caagcggagg tggaggaagt
660
ggaggtggcg gttccggcgg tggcggttca gatatacaga tgacccaatc acccagttct
720
cttagcgcgt ctgtaggcga cagggtaacc ataacctgca aggcgtccca ggacgtgtca
780
attggagttg cctggtatca gcaaaaacct gggaaagctc cgaagctcct gatttacagc
840
gcatcttacc gatatactgg tgtcccttca aggttcagtg gcagtggatc tgggacagac
900
tttacgctta ctatcagcag tctgcaacct gaggatttcg cgacctacta ctgtcagcag
960
tattacatct atccgtacac gttcggtcaa ggtacaaagg tagaaataaa acgcactttt
1020
gccattcgct ggggctttat ccctcttgtg atttacctgg gatttaagag gggtgcagat
1080
cccggaatgc ctgaaccaac tgttttgagc ctactttggg gatag
1125
<210> 39
<211> 38
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер RscFvHER2
<400> 39
aaaaaagaat tcatggaagt gcaacttgtt gagagtgg
38
<210> 40
<211> 35
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер XTOM7(noT)
<400> 40
aaaaaactcg agtccccaaa gtaggctcaa aacag
35
<210> 41
<211> 924
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность, кодирующая
scFvHER2-TOM7-myc/His
<400> 41
atggaagtgc aacttgttga gagtggcgga ggactggtcc aaccgggcgg ttcacttagg
60
ctctcatgtg cagcttcagg gttcacgttc acggactata caatggactg ggtgaggcaa
120
gcccctggta agggactgga gtgggttgct gacgttaacc ctaattccgg tgggtccatc
180
tacaaccagc gattcaaggg acgatttact ctttcagtcg acagaagcaa aaacaccctc
240
tacctccaga tgaactcctt gcgggcagag gatacagcgg tctactattg cgcgagaaac
300
ttgggaccaa gcttctactt cgactactgg gggcaaggaa cgcttgttac ggtttcaagc
360
ggaggtggag gaagtggagg tggcggttcc ggcggtggcg gttcagatat acagatgacc
420
caatcaccca gttctcttag cgcgtctgta ggcgacaggg taaccataac ctgcaaggcg
480
tcccaggacg tgtcaattgg agttgcctgg tatcagcaaa aacctgggaa agctccgaag
540
ctcctgattt acagcgcatc ttaccgatat actggtgtcc cttcaaggtt cagtggcagt
600
ggatctggga cagactttac gcttactatc agcagtctgc aacctgagga tttcgcgacc
660
tactactgtc agcagtatta catctatccg tacacgttcg gtcaaggtac aaaggtagaa
720
ataaaacgca cttttgccat tcgctggggc tttatccctc ttgtgattta cctgggattt
780
aagaggggtg cagatcccgg aatgcctgaa ccaactgttt tgagcctact ttggggactc
840
gagtctagag ggcccttcga acaaaaactc atctcagaag aggatctgaa tatgcatacc
900
ggtcatcatc accatcacca ttga
924
<210> 42
<211> 760
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность, кодирующая scFvMEL
<400> 42
ccgcggtggt acggacattg tgatgaccca gtctcaaaaa ttcatgtcca catcagtagg
60
agacagggtc agcgtcacct gcaaggccag tcagaatgtg gatactaatg tagcctggta
120
tcaacaaaaa ccagggcaat ctcctgaacc actgcttttc tcggcatcct accgttacac
180
tggagtccct gatcgcttca caggcagtgg atctgggaca gatttcactc tcaccatcag
240
caatgtgcag tctgaagact tggcagagta tttctgtcag caatataaca gctatcctct
300
gacgttcggt ggaggcacca agctggagat caaaggctcc accagcggca gcggtaagcc
360
aggctccggc gaaggcagca ccaaaggcga agtgaaggtt gaggagtctg gaggaggctt
420
ggtgcaacct ggaggatcca tgaaactctc ctgtgttgtc tctggattca ctttcggtaa
480
ttactggatg aactgggtcc gccagtctcc agagaagggg cttgagtgga ttgcagaaat
540
tagattgaaa tccaataatt ttgcaagata ttatgcggag tctgtgaaag ggaggttcac
600
catctcaaga gatgattcca aaagtagtgt ctacctgcaa atgatcaacc taagagctga
660
agatactggc atttattact gtaccagtta tggtaactac gttgggcact attttgacca
720
ctggggccaa ggcaccactc tcaccgtctc cctttgggga
760
<210> 43
<211> 1137
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность, кодирующая UB-scFvMEL-TOM7
<400> 43
atgggcagca gccatcatca tcatcatcac agcagcggcc tggtgccgcg cggcagccat
60
atgcaaatct tcgtcaaaac tctaacaggg aagactataa ccctagaggt tgaaccatcc
120
gacactattg aaaacgtcaa agctaaaatt caagataaag aaggtatccc tccggatcag
180
cagagattga tttttgctgg taagcaacta gaagatggta gaaccttgtc tgactacaac
240
atccaaaagg aatctactct tcacttggtg ttgagactcc gcggtggtac ggacattgtg
300
atgacccagt ctcaaaaatt catgtccaca tcagtaggag acagggtcag cgtcacctgc
360
aaggccagtc agaatgtgga tactaatgta gcctggtatc aacaaaaacc agggcaatct
420
cctgaaccac tgcttttctc ggcatcctac cgttacactg gagtccctga tcgcttcaca
480
ggcagtggat ctgggacaga tttcactctc accatcagca atgtgcagtc tgaagacttg
540
gcagagtatt tctgtcagca atataacagc tatcctctga cgttcggtgg aggcaccaag
600
ctggagatca aaggctccac cagcggcagc ggtaagccag gctccggcga aggcagcacc
660
aaaggcgaag tgaaggttga ggagtctgga ggaggcttgg tgcaacctgg aggatccatg
720
aaactctcct gtgttgtctc tggattcact ttcggtaatt actggatgaa ctgggtccgc
780
cagtctccag agaaggggct tgagtggatt gcagaaatta gattgaaatc caataatttt
840
gcaagatatt atgcggagtc tgtgaaaggg aggttcacca tctcaagaga tgattccaaa
900
agtagtgtct acctgcaaat gatcaaccta agagctgaag atactggcat ttattactgt
960
accagttatg gtaactacgt tgggcactat tttgaccact ggggccaagg caccactctc
1020
accgtctcct catttgccat tcgctggggc tttatccctc ttgtgattta cctgggattt
1080
aagaggggtg cagatcccgg aatgcctgaa ccaactgttt tgagcctact ttgggga
1137
<210> 44
<211> 36
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер RscFvMEL
<400> 44
aaaaaagaat tcatgaaaac aagtaaccca ggagtg
36
<210> 45
<211> 939
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность, кодирующая
scFvMEL-TOM7-myc/His
<400> 45
atgacggaca ttgtgatgac ccagtctcaa aaattcatgt ccacatcagt aggagacagg
60
gtcagcgtca cctgcaaggc cagtcagaat gtggatacta atgtagcctg gtatcaacaa
120
aaaccagggc aatctcctga accactgctt ttctcggcat cctaccgtta cactggagtc
180
cctgatcgct tcacaggcag tggatctggg acagatttca ctctcaccat cagcaatgtg
240
cagtctgaag acttggcaga gtatttctgt cagcaatata acagctatcc tctgacgttc
300
ggtggaggca ccaagctgga gatcaaaggc tccaccagcg gcagcggtaa gccaggctcc
360
ggcgaaggca gcaccaaagg cgaagtgaag gttgaggagt ctggaggagg cttggtgcaa
420
cctggaggat ccatgaaact ctcctgtgtt gtctctggat tcactttcgg taattactgg
480
atgaactggg tccgccagtc tccagagaag gggcttgagt ggattgcaga aattagattg
540
aaatccaata attttgcaag atattatgcg gagtctgtga aagggaggtt caccatctca
600
agagatgatt ccaaaagtag tgtctacctg caaatgatca acctaagagc tgaagatact
660
ggcatttatt actgtaccag ttatggtaac tacgttgggc actattttga ccactggggc
720
caaggcacca ctctcaccgt ctcctcattt gccattcgct ggggctttat ccctcttgtg
780
atttacctgg gatttaagag gggtgcagat cccggaatgc ctgaaccaac tgttttgagc
840
ctactttggg gactcgagtc tagagggccc ttcgaacaaa aactcatctc agaagaggat
900
ctgaatatgc ataccggtca tcatcaccat caccattga
939
<210> 46
<211> 750
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность, кодирующая scFvPD-L1
<400> 46
atggacatcg tgatgagcca gtctcccagc agcctggctg tgtctgctgg ggagaaggtg
60
accatgtcct gcaagagctc ccagtccctg ctgaacagcc gcaccaggaa gaactacctg
120
gcctggtacc agcagaagcc aggccagagc cccaagctcc tcatctactg ggccagcacc
180
cgggagagcg gggtgcctga ccgcttcact ggaagtggca gcggcacaga cttcaccctg
240
accatcagct ctgtgcaggc cgaggacctg gcagtgtact actgccagca aagctatgat
300
gtggtgacat ttggagctgg caccaagctg gagctgaagg gaggtggcgg aagcgggggt
360
ggaggatccg ggggcggcgg aagccaggtc caggtgcagc agagcggggc tgagctggcc
420
gagcccgggg cctctgtgaa gatgagctgc aaggcttctg gctacatctt caccagctac
480
tggatgcact ggctcaagca gaggcctggg caggggctgg agtggatcgg ctatatcaac
540
cccagcagtg actacaatga atattctgag aagttcatgg acaaagccac cctgactgct
600
gacaaggcca gcaccaccgc ctacatgcag ctgatcagcc tgacctcaga ggacagcgct
660
gtgtactact gtgcccggag cggctggctg gtgcacgggg actattattt tgattattgg
720
ggccagggca ccacactgac agtgagcagc
750
<210> 47
<211> 948
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность, кодирующая
scFvPD-L1-TOM7-myc/His
<400> 47
atggacatcg tgatgagcca gtctcccagc agcctggctg tgtctgctgg ggagaaggtg
60
accatgtcct gcaagagctc ccagtccctg ctgaacagcc gcaccaggaa gaactacctg
120
gcctggtacc agcagaagcc aggccagagc cccaagctcc tcatctactg ggccagcacc
180
cgggagagcg gggtgcctga ccgcttcact ggaagtggca gcggcacaga cttcaccctg
240
accatcagct ctgtgcaggc cgaggacctg gcagtgtact actgccagca aagctatgat
300
gtggtgacat ttggagctgg caccaagctg gagctgaagg gaggtggcgg aagcgggggt
360
ggaggatccg ggggcggcgg aagccaggtc caggtgcagc agagcggggc tgagctggcc
420
gagcccgggg cctctgtgaa gatgagctgc aaggcttctg gctacatctt caccagctac
480
tggatgcact ggctcaagca gaggcctggg caggggctgg agtggatcgg ctatatcaac
540
cccagcagtg actacaatga atattctgag aagttcatgg acaaagccac cctgactgct
600
gacaaggcca gcaccaccgc ctacatgcag ctgatcagcc tgacctcaga ggacagcgct
660
gtgtactact gtgcccggag cggctggctg gtgcacgggg actattattt tgattattgg
720
ggccagggca ccacactgac agtgagcagc ctcgagtttg ccattcgctg gggctttatc
780
cctcttgtga tttacctggg atttaagagg ggtgcagatc ccggaatgcc tgaaccaact
840
gttttgagcc tactttgggg actcgagtct agagggccct tcgaacaaaa actcatctca
900
gaagaggatc tgaatatgca taccggtcat catcaccatc accattga
948
<210> 48
<211> 395
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность p53
<400> 48
Arg Gly Gly Glu Glu Pro Gln Ser Asp Pro Ser Val Glu Pro Pro Leu
1 5 10 15
Ser Gln Glu Thr Phe Ser Asp Leu Trp Lys Leu Leu Pro Glu Asn Asn
20 25 30
Val Leu Ser Pro Leu Pro Ser Gln Ala Met Asp Asp Leu Met Leu Ser
35 40 45
Pro Asp Asp Ile Glu Gln Trp Phe Thr Glu Asp Pro Gly Pro Asp Glu
50 55 60
Ala Pro Arg Met Pro Glu Ala Ala Pro Arg Val Ala Pro Ala Pro Ala
65 70 75 80
Ala Pro Thr Pro Ala Ala Pro Ala Pro Ala Pro Ser Trp Pro Leu Ser
85 90 95
Ser Ser Val Pro Ser Gln Lys Thr Tyr Gln Gly Ser Tyr Gly Phe Arg
100 105 110
Leu Gly Phe Leu His Ser Gly Thr Ala Lys Ser Val Thr Cys Thr Tyr
115 120 125
Ser Pro Ala Leu Asn Lys Met Phe Cys Gln Leu Ala Lys Thr Cys Pro
130 135 140
Val Gln Leu Trp Val Asp Ser Thr Pro Pro Pro Gly Thr Arg Val Arg
145 150 155 160
Ala Met Ala Ile Tyr Lys Gln Ser Gln His Met Thr Glu Val Val Arg
165 170 175
Arg Cys Pro His His Glu Arg Cys Ser Asp Ser Asp Gly Leu Ala Pro
180 185 190
Pro Gln His Leu Ile Arg Val Glu Gly Asn Leu Arg Val Glu Tyr Leu
195 200 205
Asp Asp Arg Asn Thr Phe Arg His Ser Val Val Val Pro Tyr Glu Pro
210 215 220
Pro Glu Val Gly Ser Asp Cys Thr Thr Ile His Tyr Asn Tyr Met Cys
225 230 235 240
Asn Ser Ser Cys Met Gly Gly Met Asn Arg Arg Pro Ile Leu Thr Ile
245 250 255
Ile Thr Leu Glu Asp Ser Ser Gly Asn Leu Leu Gly Arg Asn Ser Phe
260 265 270
Glu Val Arg Val Cys Ala Cys Pro Gly Arg Asp Arg Arg Thr Glu Glu
275 280 285
Glu Asn Leu Arg Lys Lys Gly Glu Pro His His Glu Leu Pro Pro Gly
290 295 300
Ser Thr Lys Arg Ala Leu Pro Asn Asn Thr Ser Ser Ser Pro Gln Pro
305 310 315 320
Lys Lys Lys Pro Leu Asp Gly Glu Tyr Phe Thr Leu Gln Ile Arg Gly
325 330 335
Arg Glu Arg Phe Glu Met Phe Arg Glu Leu Asn Glu Ala Leu Glu Leu
340 345 350
Lys Asp Ala Gln Ala Gly Lys Glu Pro Gly Gly Ser Arg Ala His Ser
355 360 365
Ser His Leu Lys Ser Lys Lys Gly Gln Ser Thr Ser Arg His Lys Lys
370 375 380
Leu Met Phe Lys Thr Glu Gly Pro Asp Ser Asp
385 390 395
<210> 49
<211> 488
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность UB-p53
<400> 49
Met Gly Ser Ser His His His His His His Ser Ser Gly Leu Val Pro
1 5 10 15
Arg Gly Ser His Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr
20 25 30
Ile Thr Leu Glu Val Glu Pro Ser Asp Thr Ile Glu Asn Val Lys Ala
35 40 45
Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile
50 55 60
Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu Ser Asp Tyr Asn
65 70 75 80
Ile Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly
85 90 95
Glu Glu Pro Gln Ser Asp Pro Ser Val Glu Pro Pro Leu Ser Gln Glu
100 105 110
Thr Phe Ser Asp Leu Trp Lys Leu Leu Pro Glu Asn Asn Val Leu Ser
115 120 125
Pro Leu Pro Ser Gln Ala Met Asp Asp Leu Met Leu Ser Pro Asp Asp
130 135 140
Ile Glu Gln Trp Phe Thr Glu Asp Pro Gly Pro Asp Glu Ala Pro Arg
145 150 155 160
Met Pro Glu Ala Ala Pro Arg Val Ala Pro Ala Pro Ala Ala Pro Thr
165 170 175
Pro Ala Ala Pro Ala Pro Ala Pro Ser Trp Pro Leu Ser Ser Ser Val
180 185 190
Pro Ser Gln Lys Thr Tyr Gln Gly Ser Tyr Gly Phe Arg Leu Gly Phe
195 200 205
Leu His Ser Gly Thr Ala Lys Ser Val Thr Cys Thr Tyr Ser Pro Ala
210 215 220
Leu Asn Lys Met Phe Cys Gln Leu Ala Lys Thr Cys Pro Val Gln Leu
225 230 235 240
Trp Val Asp Ser Thr Pro Pro Pro Gly Thr Arg Val Arg Ala Met Ala
245 250 255
Ile Tyr Lys Gln Ser Gln His Met Thr Glu Val Val Arg Arg Cys Pro
260 265 270
His His Glu Arg Cys Ser Asp Ser Asp Gly Leu Ala Pro Pro Gln His
275 280 285
Leu Ile Arg Val Glu Gly Asn Leu Arg Val Glu Tyr Leu Asp Asp Arg
290 295 300
Asn Thr Phe Arg His Ser Val Val Val Pro Tyr Glu Pro Pro Glu Val
305 310 315 320
Gly Ser Asp Cys Thr Thr Ile His Tyr Asn Tyr Met Cys Asn Ser Ser
325 330 335
Cys Met Gly Gly Met Asn Arg Arg Pro Ile Leu Thr Ile Ile Thr Leu
340 345 350
Glu Asp Ser Ser Gly Asn Leu Leu Gly Arg Asn Ser Phe Glu Val Arg
355 360 365
Val Cys Ala Cys Pro Gly Arg Asp Arg Arg Thr Glu Glu Glu Asn Leu
370 375 380
Arg Lys Lys Gly Glu Pro His His Glu Leu Pro Pro Gly Ser Thr Lys
385 390 395 400
Arg Ala Leu Pro Asn Asn Thr Ser Ser Ser Pro Gln Pro Lys Lys Lys
405 410 415
Pro Leu Asp Gly Glu Tyr Phe Thr Leu Gln Ile Arg Gly Arg Glu Arg
420 425 430
Phe Glu Met Phe Arg Glu Leu Asn Glu Ala Leu Glu Leu Lys Asp Ala
435 440 445
Gln Ala Gly Lys Glu Pro Gly Gly Ser Arg Ala His Ser Ser His Leu
450 455 460
Lys Ser Lys Lys Gly Gln Ser Thr Ser Arg His Lys Lys Leu Met Phe
465 470 475 480
Lys Thr Glu Gly Pro Asp Ser Asp
485
<210> 50
<211> 504
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность TOM70-UB-p53
<400> 50
Met Lys Ser Phe Ile Thr Arg Asn Lys Thr Ala Ile Phe Ala Thr Val
1 5 10 15
Ala Ala Thr Gly Thr Ala Ile Gly Ala Tyr Tyr Tyr Tyr Gln Ile Phe
20 25 30
Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Pro Ser
35 40 45
Asp Thr Ile Glu Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile
50 55 60
Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp
65 70 75 80
Gly Arg Thr Leu Ser Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His
85 90 95
Leu Val Leu Arg Leu Arg Gly Gly Glu Glu Pro Gln Ser Asp Pro Ser
100 105 110
Val Glu Pro Pro Leu Ser Gln Glu Thr Phe Ser Asp Leu Trp Lys Leu
115 120 125
Leu Pro Glu Asn Asn Val Leu Ser Pro Leu Pro Ser Gln Ala Met Asp
130 135 140
Asp Leu Met Leu Ser Pro Asp Asp Ile Glu Gln Trp Phe Thr Glu Asp
145 150 155 160
Pro Gly Pro Asp Glu Ala Pro Arg Met Pro Glu Ala Ala Pro Arg Val
165 170 175
Ala Pro Ala Pro Ala Ala Pro Thr Pro Ala Ala Pro Ala Pro Ala Pro
180 185 190
Ser Trp Pro Leu Ser Ser Ser Val Pro Ser Gln Lys Thr Tyr Gln Gly
195 200 205
Ser Tyr Gly Phe Arg Leu Gly Phe Leu His Ser Gly Thr Ala Lys Ser
210 215 220
Val Thr Cys Thr Tyr Ser Pro Ala Leu Asn Lys Met Phe Cys Gln Leu
225 230 235 240
Ala Lys Thr Cys Pro Val Gln Leu Trp Val Asp Ser Thr Pro Pro Pro
245 250 255
Gly Thr Arg Val Arg Ala Met Ala Ile Tyr Lys Gln Ser Gln His Met
260 265 270
Thr Glu Val Val Arg Arg Cys Pro His His Glu Arg Cys Ser Asp Ser
275 280 285
Asp Gly Leu Ala Pro Pro Gln His Leu Ile Arg Val Glu Gly Asn Leu
290 295 300
Arg Val Glu Tyr Leu Asp Asp Arg Asn Thr Phe Arg His Ser Val Val
305 310 315 320
Val Pro Tyr Glu Pro Pro Glu Val Gly Ser Asp Cys Thr Thr Ile His
325 330 335
Tyr Asn Tyr Met Cys Asn Ser Ser Cys Met Gly Gly Met Asn Arg Arg
340 345 350
Pro Ile Leu Thr Ile Ile Thr Leu Glu Asp Ser Ser Gly Asn Leu Leu
355 360 365
Gly Arg Asn Ser Phe Glu Val Arg Val Cys Ala Cys Pro Gly Arg Asp
370 375 380
Arg Arg Thr Glu Glu Glu Asn Leu Arg Lys Lys Gly Glu Pro His His
385 390 395 400
Glu Leu Pro Pro Gly Ser Thr Lys Arg Ala Leu Pro Asn Asn Thr Ser
405 410 415
Ser Ser Pro Gln Pro Lys Lys Lys Pro Leu Asp Gly Glu Tyr Phe Thr
420 425 430
Leu Gln Ile Arg Gly Arg Glu Arg Phe Glu Met Phe Arg Glu Leu Asn
435 440 445
Glu Ala Leu Glu Leu Lys Asp Ala Gln Ala Gly Lys Glu Pro Gly Gly
450 455 460
Ser Arg Ala His Ser Ser His Leu Lys Ser Lys Lys Gly Gln Ser Thr
465 470 475 480
Ser Arg His Lys Lys Leu Met Phe Lys Thr Glu Gly Pro Asp Ser Asp
485 490 495
Leu Glu His His His His His His
500
<210> 51
<211> 519
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность TOM70-(GGGGS)3-UB-p53
<400> 51
Met Lys Ser Phe Ile Thr Arg Asn Lys Thr Ala Ile Phe Ala Thr Val
1 5 10 15
Ala Ala Thr Gly Thr Ala Ile Gly Ala Tyr Tyr Tyr Tyr Gly Gly Gly
20 25 30
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ile Phe Val
35 40 45
Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Pro Ser Asp
50 55 60
Thr Ile Glu Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile Pro
65 70 75 80
Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp Gly
85 90 95
Arg Thr Leu Ser Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His Leu
100 105 110
Val Leu Arg Leu Arg Gly Gly Glu Glu Pro Gln Ser Asp Pro Ser Val
115 120 125
Glu Pro Pro Leu Ser Gln Glu Thr Phe Ser Asp Leu Trp Lys Leu Leu
130 135 140
Pro Glu Asn Asn Val Leu Ser Pro Leu Pro Ser Gln Ala Met Asp Asp
145 150 155 160
Leu Met Leu Ser Pro Asp Asp Ile Glu Gln Trp Phe Thr Glu Asp Pro
165 170 175
Gly Pro Asp Glu Ala Pro Arg Met Pro Glu Ala Ala Pro Arg Val Ala
180 185 190
Pro Ala Pro Ala Ala Pro Thr Pro Ala Ala Pro Ala Pro Ala Pro Ser
195 200 205
Trp Pro Leu Ser Ser Ser Val Pro Ser Gln Lys Thr Tyr Gln Gly Ser
210 215 220
Tyr Gly Phe Arg Leu Gly Phe Leu His Ser Gly Thr Ala Lys Ser Val
225 230 235 240
Thr Cys Thr Tyr Ser Pro Ala Leu Asn Lys Met Phe Cys Gln Leu Ala
245 250 255
Lys Thr Cys Pro Val Gln Leu Trp Val Asp Ser Thr Pro Pro Pro Gly
260 265 270
Thr Arg Val Arg Ala Met Ala Ile Tyr Lys Gln Ser Gln His Met Thr
275 280 285
Glu Val Val Arg Arg Cys Pro His His Glu Arg Cys Ser Asp Ser Asp
290 295 300
Gly Leu Ala Pro Pro Gln His Leu Ile Arg Val Glu Gly Asn Leu Arg
305 310 315 320
Val Glu Tyr Leu Asp Asp Arg Asn Thr Phe Arg His Ser Val Val Val
325 330 335
Pro Tyr Glu Pro Pro Glu Val Gly Ser Asp Cys Thr Thr Ile His Tyr
340 345 350
Asn Tyr Met Cys Asn Ser Ser Cys Met Gly Gly Met Asn Arg Arg Pro
355 360 365
Ile Leu Thr Ile Ile Thr Leu Glu Asp Ser Ser Gly Asn Leu Leu Gly
370 375 380
Arg Asn Ser Phe Glu Val Arg Val Cys Ala Cys Pro Gly Arg Asp Arg
385 390 395 400
Arg Thr Glu Glu Glu Asn Leu Arg Lys Lys Gly Glu Pro His His Glu
405 410 415
Leu Pro Pro Gly Ser Thr Lys Arg Ala Leu Pro Asn Asn Thr Ser Ser
420 425 430
Ser Pro Gln Pro Lys Lys Lys Pro Leu Asp Gly Glu Tyr Phe Thr Leu
435 440 445
Gln Ile Arg Gly Arg Glu Arg Phe Glu Met Phe Arg Glu Leu Asn Glu
450 455 460
Ala Leu Glu Leu Lys Asp Ala Gln Ala Gly Lys Glu Pro Gly Gly Ser
465 470 475 480
Arg Ala His Ser Ser His Leu Lys Ser Lys Lys Gly Gln Ser Thr Ser
485 490 495
Arg His Lys Lys Leu Met Phe Lys Thr Glu Gly Pro Asp Ser Asp Leu
500 505 510
Glu His His His His His His
515
<210> 52
<211> 444
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность TOM70-(GGGGS)3-p53
<400> 52
Met Lys Ser Phe Ile Thr Arg Asn Lys Thr Ala Ile Phe Ala Thr Val
1 5 10 15
Ala Ala Thr Gly Thr Ala Ile Gly Ala Tyr Tyr Tyr Tyr Gly Gly Gly
20 25 30
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Glu Pro Gln
35 40 45
Ser Asp Pro Ser Val Glu Pro Pro Leu Ser Gln Glu Thr Phe Ser Asp
50 55 60
Leu Trp Lys Leu Leu Pro Glu Asn Asn Val Leu Ser Pro Leu Pro Ser
65 70 75 80
Gln Ala Met Asp Asp Leu Met Leu Ser Pro Asp Asp Ile Glu Gln Trp
85 90 95
Phe Thr Glu Asp Pro Gly Pro Asp Glu Ala Pro Arg Met Pro Glu Ala
100 105 110
Ala Pro Arg Val Ala Pro Ala Pro Ala Ala Pro Thr Pro Ala Ala Pro
115 120 125
Ala Pro Ala Pro Ser Trp Pro Leu Ser Ser Ser Val Pro Ser Gln Lys
130 135 140
Thr Tyr Gln Gly Ser Tyr Gly Phe Arg Leu Gly Phe Leu His Ser Gly
145 150 155 160
Thr Ala Lys Ser Val Thr Cys Thr Tyr Ser Pro Ala Leu Asn Lys Met
165 170 175
Phe Cys Gln Leu Ala Lys Thr Cys Pro Val Gln Leu Trp Val Asp Ser
180 185 190
Thr Pro Pro Pro Gly Thr Arg Val Arg Ala Met Ala Ile Tyr Lys Gln
195 200 205
Ser Gln His Met Thr Glu Val Val Arg Arg Cys Pro His His Glu Arg
210 215 220
Cys Ser Asp Ser Asp Gly Leu Ala Pro Pro Gln His Leu Ile Arg Val
225 230 235 240
Glu Gly Asn Leu Arg Val Glu Tyr Leu Asp Asp Arg Asn Thr Phe Arg
245 250 255
His Ser Val Val Val Pro Tyr Glu Pro Pro Glu Val Gly Ser Asp Cys
260 265 270
Thr Thr Ile His Tyr Asn Tyr Met Cys Asn Ser Ser Cys Met Gly Gly
275 280 285
Met Asn Arg Arg Pro Ile Leu Thr Ile Ile Thr Leu Glu Asp Ser Ser
290 295 300
Gly Asn Leu Leu Gly Arg Asn Ser Phe Glu Val Arg Val Cys Ala Cys
305 310 315 320
Pro Gly Arg Asp Arg Arg Thr Glu Glu Glu Asn Leu Arg Lys Lys Gly
325 330 335
Glu Pro His His Glu Leu Pro Pro Gly Ser Thr Lys Arg Ala Leu Pro
340 345 350
Asn Asn Thr Ser Ser Ser Pro Gln Pro Lys Lys Lys Pro Leu Asp Gly
355 360 365
Glu Tyr Phe Thr Leu Gln Ile Arg Gly Arg Glu Arg Phe Glu Met Phe
370 375 380
Arg Glu Leu Asn Glu Ala Leu Glu Leu Lys Asp Ala Gln Ala Gly Lys
385 390 395 400
Glu Pro Gly Gly Ser Arg Ala His Ser Ser His Leu Lys Ser Lys Lys
405 410 415
Gly Gln Ser Thr Ser Arg His Lys Lys Leu Met Phe Lys Thr Glu Gly
420 425 430
Pro Asp Ser Asp Leu Glu His His His His His His
435 440
<210> 53
<211> 525
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность UB-p53-TOM7
<400> 53
Met Gly Ser Ser His His His His His His Ser Ser Gly Leu Val Pro
1 5 10 15
Arg Gly Ser His Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr
20 25 30
Ile Thr Leu Glu Val Glu Pro Ser Asp Thr Ile Glu Asn Val Lys Ala
35 40 45
Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile
50 55 60
Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu Ser Asp Tyr Asn
65 70 75 80
Ile Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly
85 90 95
Glu Glu Pro Gln Ser Asp Pro Ser Val Glu Pro Pro Leu Ser Gln Glu
100 105 110
Thr Phe Ser Asp Leu Trp Lys Leu Leu Pro Glu Asn Asn Val Leu Ser
115 120 125
Pro Leu Pro Ser Gln Ala Met Asp Asp Leu Met Leu Ser Pro Asp Asp
130 135 140
Ile Glu Gln Trp Phe Thr Glu Asp Pro Gly Pro Asp Glu Ala Pro Arg
145 150 155 160
Met Pro Glu Ala Ala Pro Arg Val Ala Pro Ala Pro Ala Ala Pro Thr
165 170 175
Pro Ala Ala Pro Ala Pro Ala Pro Ser Trp Pro Leu Ser Ser Ser Val
180 185 190
Pro Ser Gln Lys Thr Tyr Gln Gly Ser Tyr Gly Phe Arg Leu Gly Phe
195 200 205
Leu His Ser Gly Thr Ala Lys Ser Val Thr Cys Thr Tyr Ser Pro Ala
210 215 220
Leu Asn Lys Met Phe Cys Gln Leu Ala Lys Thr Cys Pro Val Gln Leu
225 230 235 240
Trp Val Asp Ser Thr Pro Pro Pro Gly Thr Arg Val Arg Ala Met Ala
245 250 255
Ile Tyr Lys Gln Ser Gln His Met Thr Glu Val Val Arg Arg Cys Pro
260 265 270
His His Glu Arg Cys Ser Asp Ser Asp Gly Leu Ala Pro Pro Gln His
275 280 285
Leu Ile Arg Val Glu Gly Asn Leu Arg Val Glu Tyr Leu Asp Asp Arg
290 295 300
Asn Thr Phe Arg His Ser Val Val Val Pro Tyr Glu Pro Pro Glu Val
305 310 315 320
Gly Ser Asp Cys Thr Thr Ile His Tyr Asn Tyr Met Cys Asn Ser Ser
325 330 335
Cys Met Gly Gly Met Asn Arg Arg Pro Ile Leu Thr Ile Ile Thr Leu
340 345 350
Glu Asp Ser Ser Gly Asn Leu Leu Gly Arg Asn Ser Phe Glu Val His
355 360 365
Val Cys Ala Cys Pro Gly Arg Asp Arg Arg Thr Glu Glu Glu Asn Leu
370 375 380
Arg Lys Lys Gly Glu Pro His His Glu Leu Pro Pro Gly Ser Thr Lys
385 390 395 400
Arg Ala Leu Ser Asn Asn Thr Ser Ser Ser Pro Gln Pro Lys Lys Lys
405 410 415
Pro Leu Asp Gly Glu Tyr Phe Thr Leu Gln Ile Arg Gly Arg Glu Arg
420 425 430
Phe Glu Met Phe Arg Glu Leu Asn Glu Ala Leu Glu Leu Lys Asp Ala
435 440 445
Gln Ala Gly Lys Glu Pro Gly Gly Ser Arg Ala His Ser Ser His Leu
450 455 460
Lys Ser Lys Lys Gly Gln Ser Thr Ser Arg His Lys Lys Leu Met Phe
465 470 475 480
Lys Thr Glu Gly Pro Asp Ser Asp Leu Glu Phe Ala Ile Arg Trp Gly
485 490 495
Phe Ile Pro Leu Val Ile Tyr Leu Gly Phe Lys Arg Gly Ala Asp Pro
500 505 510
Gly Met Pro Glu Pro Thr Val Leu Ser Leu Leu Trp Gly
515 520 525
<210> 54
<211> 421
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность p53-myc/His
<400> 54
Met Glu Glu Pro Gln Ser Asp Pro Ser Val Glu Pro Pro Leu Ser Gln
1 5 10 15
Glu Thr Phe Ser Asp Leu Trp Lys Leu Leu Pro Glu Asn Asn Val Leu
20 25 30
Ser Pro Leu Pro Ser Gln Ala Met Asp Asp Leu Met Leu Ser Pro Asp
35 40 45
Asp Ile Glu Gln Trp Phe Thr Glu Asp Pro Gly Pro Asp Glu Ala Pro
50 55 60
Arg Met Pro Glu Ala Ala Pro Arg Val Ala Pro Ala Pro Ala Ala Pro
65 70 75 80
Thr Pro Ala Ala Pro Ala Pro Ala Pro Ser Trp Pro Leu Ser Ser Ser
85 90 95
Val Pro Ser Gln Lys Thr Tyr Gln Gly Ser Tyr Gly Phe Arg Leu Gly
100 105 110
Phe Leu His Ser Gly Thr Ala Lys Ser Val Thr Cys Thr Tyr Ser Pro
115 120 125
Ala Leu Asn Lys Met Phe Cys Gln Leu Ala Lys Thr Cys Pro Val Gln
130 135 140
Leu Trp Val Asp Ser Thr Pro Pro Pro Gly Thr Arg Val Arg Ala Met
145 150 155 160
Ala Ile Tyr Lys Gln Ser Gln His Met Thr Glu Val Val Arg Arg Cys
165 170 175
Pro His His Glu Arg Cys Ser Asp Ser Asp Gly Leu Ala Pro Pro Gln
180 185 190
His Leu Ile Arg Val Glu Gly Asn Leu Arg Val Glu Tyr Leu Asp Asp
195 200 205
Arg Asn Thr Phe Arg His Ser Val Val Val Pro Tyr Glu Pro Pro Glu
210 215 220
Val Gly Ser Asp Cys Thr Thr Ile His Tyr Asn Tyr Met Cys Asn Ser
225 230 235 240
Ser Cys Met Gly Gly Met Asn Arg Arg Pro Ile Leu Thr Ile Ile Thr
245 250 255
Leu Glu Asp Ser Ser Gly Asn Leu Leu Gly Arg Asn Ser Phe Glu Val
260 265 270
Arg Val Cys Ala Cys Pro Gly Arg Asp Arg Arg Thr Glu Glu Glu Asn
275 280 285
Leu Arg Lys Lys Gly Glu Pro His His Glu Leu Pro Pro Gly Ser Thr
290 295 300
Lys Arg Ala Leu Pro Asn Asn Thr Ser Ser Ser Pro Gln Pro Lys Lys
305 310 315 320
Lys Pro Leu Asp Gly Glu Tyr Phe Thr Leu Gln Ile Arg Gly Arg Glu
325 330 335
Arg Phe Glu Met Phe Arg Glu Leu Asn Glu Ala Leu Glu Leu Lys Asp
340 345 350
Ala Gln Ala Gly Lys Glu Pro Gly Gly Ser Arg Ala His Ser Ser His
355 360 365
Leu Lys Ser Lys Lys Gly Gln Ser Thr Ser Arg His Lys Lys Leu Met
370 375 380
Phe Lys Thr Glu Gly Pro Asp Ser Asp Leu Glu Ser Arg Gly Pro Phe
385 390 395 400
Glu Gln Lys Leu Ile Ser Glu Glu Asp Leu Asn Met His Thr Gly His
405 410 415
His His His His His
420
<210> 55
<211> 230
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность гранзима B
<400> 55
Arg Gly Gly Ile Ile Gly Gly His Glu Ala Lys Pro His Ser Arg Pro
1 5 10 15
Tyr Met Ala Tyr Leu Met Ile Trp Asp Gln Lys Ser Leu Lys Arg Cys
20 25 30
Gly Gly Phe Leu Ile Gln Asp Asp Phe Val Leu Thr Ala Ala His Cys
35 40 45
Trp Gly Ser Ser Ile Asn Val Thr Leu Gly Ala His Asn Ile Lys Glu
50 55 60
Gln Glu Pro Thr Gln Gln Phe Ile Pro Val Lys Arg Pro Ile Pro His
65 70 75 80
Pro Ala Tyr Asn Pro Lys Asn Phe Ser Asn Asp Ile Met Leu Leu Gln
85 90 95
Leu Glu Arg Lys Ala Lys Arg Thr Arg Ala Val Gln Pro Leu Arg Leu
100 105 110
Pro Ser Asn Lys Ala Gln Val Lys Pro Gly Gln Thr Cys Ser Val Ala
115 120 125
Gly Trp Gly Gln Thr Ala Pro Leu Gly Lys His Ser His Thr Leu Gln
130 135 140
Glu Val Lys Met Thr Val Gln Glu Asp Arg Lys Cys Glu Ser Asp Leu
145 150 155 160
Arg His Tyr Tyr Asp Ser Thr Ile Glu Leu Cys Val Gly Asp Pro Glu
165 170 175
Ile Lys Lys Thr Ser Phe Lys Gly Asp Ser Gly Gly Pro Leu Val Cys
180 185 190
Asn Lys Val Ala Gln Gly Ile Val Ser Tyr Gly Arg Asn Asn Gly Met
195 200 205
Pro Pro Arg Ala Cys Thr Lys Val Ser Ser Phe Val His Trp Ile Lys
210 215 220
Lys Thr Met Lys Arg Tyr
225 230
<210> 56
<211> 354
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность TOM70-(GGGGS)3-UB-гранзим
B
<400> 56
Met Lys Ser Phe Ile Thr Arg Asn Lys Thr Ala Ile Phe Ala Thr Val
1 5 10 15
Ala Ala Thr Gly Thr Ala Ile Gly Ala Tyr Tyr Tyr Tyr Gly Gly Gly
20 25 30
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ile Phe Val
35 40 45
Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Pro Ser Asp
50 55 60
Thr Ile Glu Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile Pro
65 70 75 80
Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp Gly
85 90 95
Arg Thr Leu Ser Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His Leu
100 105 110
Val Leu Arg Leu Arg Gly Gly Ile Ile Gly Gly His Glu Ala Lys Pro
115 120 125
His Ser Arg Pro Tyr Met Ala Tyr Leu Met Ile Trp Asp Gln Lys Ser
130 135 140
Leu Lys Arg Cys Gly Gly Phe Leu Ile Arg Asp Asp Phe Val Leu Thr
145 150 155 160
Ala Ala His Cys Trp Gly Ser Ser Ile Asn Val Thr Leu Gly Ala His
165 170 175
Asn Ile Lys Glu Gln Glu Pro Thr Gln Gln Phe Ile Pro Val Lys Arg
180 185 190
Pro Ile Pro His Pro Ala Tyr Asn Pro Lys Asn Phe Ser Asn Asp Ile
195 200 205
Met Leu Leu Gln Leu Glu Arg Lys Ala Lys Arg Thr Arg Ala Val Gln
210 215 220
Pro Leu Arg Leu Pro Ser Asn Lys Ala Gln Val Lys Pro Gly Gln Thr
225 230 235 240
Cys Ser Val Ala Gly Trp Gly Gln Thr Ala Pro Leu Gly Lys His Ser
245 250 255
His Thr Leu Gln Glu Val Lys Met Thr Val Gln Glu Asp Arg Lys Cys
260 265 270
Glu Ser Asp Leu Arg His Tyr Tyr Asp Ser Thr Ile Glu Leu Cys Val
275 280 285
Gly Asp Pro Glu Ile Lys Lys Thr Ser Phe Lys Gly Asp Ser Gly Gly
290 295 300
Pro Leu Val Cys Asn Lys Val Ala Gln Gly Ile Val Ser Tyr Gly Arg
305 310 315 320
Asn Asn Gly Met Pro Pro Arg Ala Cys Thr Lys Val Ser Ser Phe Val
325 330 335
His Trp Ile Lys Lys Thr Met Lys Arg Tyr Leu Glu His His His His
340 345 350
His His
<210> 57
<211> 360
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность UB-гранзим B-TOM7
<400> 57
Met Gly Ser Ser His His His His His His Ser Ser Gly Leu Val Pro
1 5 10 15
Arg Gly Ser His Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr
20 25 30
Ile Thr Leu Glu Val Glu Pro Ser Asp Thr Ile Glu Asn Val Lys Ala
35 40 45
Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile
50 55 60
Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu Ser Asp Tyr Asn
65 70 75 80
Ile Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly
85 90 95
Ile Ile Gly Gly His Glu Ala Lys Pro His Ser Arg Pro Tyr Met Ala
100 105 110
Tyr Leu Met Ile Trp Asp Gln Lys Ser Leu Lys Arg Cys Gly Gly Phe
115 120 125
Leu Ile Arg Asp Asp Phe Val Leu Thr Ala Ala His Cys Trp Gly Ser
130 135 140
Ser Ile Asn Val Thr Leu Gly Ala His Asn Ile Lys Glu Gln Glu Pro
145 150 155 160
Thr Gln Gln Phe Ile Pro Val Lys Arg Pro Ile Pro His Pro Ala Tyr
165 170 175
Asn Pro Lys Asn Phe Ser Asn Asp Ile Met Leu Leu Gln Leu Glu Arg
180 185 190
Lys Ala Lys Arg Thr Arg Ala Val Gln Pro Leu Arg Leu Pro Ser Asn
195 200 205
Lys Ala Gln Val Lys Pro Gly Gln Thr Cys Ser Val Ala Gly Trp Gly
210 215 220
Gln Thr Ala Pro Leu Gly Lys His Ser His Thr Leu Gln Glu Val Lys
225 230 235 240
Met Thr Val Gln Glu Asp Arg Lys Cys Glu Ser Asp Leu Arg His Tyr
245 250 255
Tyr Asp Ser Thr Ile Glu Leu Cys Val Gly Asp Pro Glu Ile Lys Lys
260 265 270
Thr Ser Phe Lys Gly Asp Ser Gly Gly Pro Leu Val Cys Asn Lys Val
275 280 285
Ala Gln Gly Ile Val Ser Tyr Gly Arg Asn Asn Gly Met Pro Pro Arg
290 295 300
Ala Cys Thr Lys Val Ser Ser Phe Val His Trp Ile Lys Lys Thr Met
305 310 315 320
Lys Arg Tyr Leu Glu Phe Ala Ile Arg Trp Gly Phe Ile Pro Leu Val
325 330 335
Ile Tyr Leu Gly Phe Lys Arg Gly Ala Asp Pro Gly Met Pro Glu Pro
340 345 350
Thr Val Leu Ser Leu Leu Trp Gly
355 360
<210> 58
<211> 189
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность RKIP
<400> 58
Arg Gly Gly Pro Val Asp Leu Ser Lys Trp Ser Gly Pro Leu Ser Leu
1 5 10 15
Gln Glu Val Asp Glu Gln Pro Gln His Pro Leu His Val Thr Tyr Ala
20 25 30
Gly Ala Ala Val Asp Glu Leu Gly Lys Val Leu Thr Pro Thr Gln Val
35 40 45
Lys Asn Arg Pro Thr Ser Ile Ser Trp Asp Gly Leu Asp Ser Gly Lys
50 55 60
Leu Tyr Thr Leu Val Leu Thr Asp Pro Asp Ala Pro Ser Arg Lys Asp
65 70 75 80
Pro Lys Tyr Arg Glu Trp His His Phe Leu Val Val Asn Met Lys Gly
85 90 95
Asn Asp Ile Ser Ser Gly Thr Val Leu Ser Asp Tyr Val Gly Ser Gly
100 105 110
Pro Pro Lys Gly Thr Gly Leu His Arg Tyr Val Trp Leu Val Tyr Glu
115 120 125
Gln Asp Arg Pro Leu Lys Cys Asp Glu Pro Ile Leu Ser Asn Arg Ser
130 135 140
Gly Asp His Arg Gly Lys Phe Lys Val Ala Ser Phe Arg Lys Lys Tyr
145 150 155 160
Glu Leu Arg Ala Pro Val Ala Gly Thr Cys Tyr Gln Ala Glu Trp Asp
165 170 175
Asp Tyr Val Pro Lys Leu Tyr Glu Gln Leu Ser Gly Lys
180 185
<210> 59
<211> 313
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность TOM70-(GGGGS)3-UB-RKIP
<400> 59
Met Lys Ser Phe Ile Thr Arg Asn Lys Thr Ala Ile Phe Ala Thr Val
1 5 10 15
Ala Ala Thr Gly Thr Ala Ile Gly Ala Tyr Tyr Tyr Tyr Gly Gly Gly
20 25 30
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ile Phe Val
35 40 45
Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Pro Ser Asp
50 55 60
Thr Ile Glu Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile Pro
65 70 75 80
Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp Gly
85 90 95
Arg Thr Leu Ser Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His Leu
100 105 110
Val Leu Arg Leu Arg Gly Gly Pro Val Asp Leu Ser Lys Trp Ser Gly
115 120 125
Pro Leu Ser Leu Gln Glu Val Asp Glu Gln Pro Gln His Pro Leu His
130 135 140
Val Thr Tyr Ala Gly Ala Ala Val Asp Glu Leu Gly Lys Val Leu Thr
145 150 155 160
Pro Thr Gln Val Lys Asn Arg Pro Thr Ser Ile Ser Trp Asp Gly Leu
165 170 175
Asp Ser Gly Lys Leu Tyr Thr Leu Val Leu Thr Asp Pro Asp Ala Pro
180 185 190
Ser Arg Lys Asp Pro Lys Tyr Arg Glu Trp His His Phe Leu Val Val
195 200 205
Asn Met Lys Gly Asn Asp Ile Ser Ser Gly Thr Val Leu Ser Asp Tyr
210 215 220
Val Gly Ser Gly Pro Pro Lys Gly Thr Gly Leu His Arg Tyr Val Trp
225 230 235 240
Leu Val Tyr Glu Gln Asp Arg Pro Leu Lys Cys Asp Glu Pro Ile Leu
245 250 255
Ser Asn Arg Ser Gly Asp His Arg Gly Lys Phe Lys Val Ala Ser Phe
260 265 270
Arg Lys Lys Tyr Glu Leu Arg Ala Pro Val Ala Gly Thr Cys Tyr Gln
275 280 285
Ala Glu Trp Asp Asp Tyr Val Pro Lys Leu Tyr Glu Gln Leu Ser Gly
290 295 300
Lys Leu Glu His His His His His His
305 310
<210> 60
<211> 405
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность PTEN
<400> 60
Arg Gly Gly Thr Ala Ile Ile Lys Glu Ile Val Ser Arg Asn Lys Arg
1 5 10 15
Arg Tyr Gln Glu Asp Gly Phe Asp Leu Asp Leu Thr Tyr Ile Tyr Pro
20 25 30
Asn Ile Ile Ala Met Gly Phe Pro Ala Glu Arg Leu Glu Gly Val Tyr
35 40 45
Arg Asn Asn Ile Asp Asp Val Val Arg Phe Leu Asp Ser Lys His Lys
50 55 60
Asn His Tyr Lys Ile Tyr Asn Leu Cys Ala Glu Arg His Tyr Asp Thr
65 70 75 80
Ala Lys Phe Asn Cys Arg Val Ala Gln Tyr Pro Phe Glu Asp His Asn
85 90 95
Pro Pro Gln Leu Glu Leu Ile Lys Pro Phe Cys Glu Asp Leu Asp Gln
100 105 110
Trp Leu Ser Glu Asp Asp Asn His Val Ala Ala Ile His Cys Lys Ala
115 120 125
Gly Lys Gly Arg Thr Gly Val Met Ile Cys Ala Tyr Leu Leu His Arg
130 135 140
Gly Lys Phe Leu Lys Ala Gln Glu Ala Leu Asp Phe Tyr Gly Glu Val
145 150 155 160
Arg Thr Arg Asp Lys Lys Gly Val Thr Ile Pro Ser Gln Arg Arg Tyr
165 170 175
Val Tyr Tyr Tyr Ser Tyr Leu Leu Lys Asn His Leu Asp Tyr Arg Pro
180 185 190
Val Ala Leu Leu Phe His Lys Met Met Phe Glu Thr Ile Pro Met Phe
195 200 205
Ser Gly Gly Thr Cys Asn Pro Gln Phe Val Val Cys Gln Leu Lys Val
210 215 220
Lys Ile Tyr Ser Ser Asn Ser Gly Pro Thr Arg Arg Glu Asp Lys Phe
225 230 235 240
Met Tyr Phe Glu Phe Pro Gln Pro Leu Pro Val Cys Gly Asp Ile Lys
245 250 255
Val Glu Phe Phe His Lys Gln Asn Lys Met Leu Lys Lys Asp Lys Met
260 265 270
Phe His Phe Trp Val Asn Thr Phe Phe Ile Pro Gly Pro Glu Glu Thr
275 280 285
Ser Glu Lys Val Glu Asn Gly Ser Leu Cys Asp Gln Glu Ile Asp Ser
290 295 300
Ile Cys Ser Ile Glu Arg Ala Asp Asn Asp Lys Glu Tyr Leu Val Leu
305 310 315 320
Thr Leu Thr Lys Asn Asp Leu Asp Lys Ala Asn Lys Asp Lys Ala Asn
325 330 335
Arg Tyr Phe Ser Pro Asn Phe Lys Val Lys Leu Tyr Phe Thr Lys Thr
340 345 350
Val Glu Glu Pro Ser Asn Pro Glu Ala Ser Ser Ser Thr Ser Val Thr
355 360 365
Pro Asp Val Ser Asp Asn Glu Pro Asp His Tyr Arg Tyr Ser Asp Thr
370 375 380
Thr Asp Ser Asp Pro Glu Asn Glu Pro Phe Asp Glu Asp Gln His Thr
385 390 395 400
Gln Ile Thr Lys Val
405
<210> 61
<211> 529
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность TOM70-(GGGGS)3-UB-PTEN
<400> 61
Met Lys Ser Phe Ile Thr Arg Asn Lys Thr Ala Ile Phe Ala Thr Val
1 5 10 15
Ala Ala Thr Gly Thr Ala Ile Gly Ala Tyr Tyr Tyr Tyr Gly Gly Gly
20 25 30
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ile Phe Val
35 40 45
Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Pro Ser Asp
50 55 60
Thr Ile Glu Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile Pro
65 70 75 80
Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp Gly
85 90 95
Arg Thr Leu Ser Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His Leu
100 105 110
Val Leu Arg Leu Arg Gly Gly Thr Ala Ile Ile Lys Glu Ile Val Ser
115 120 125
Arg Asn Lys Arg Arg Tyr Gln Glu Asp Gly Phe Asp Leu Asp Leu Thr
130 135 140
Tyr Ile Tyr Pro Asn Ile Ile Ala Met Gly Phe Pro Ala Glu Arg Leu
145 150 155 160
Glu Gly Val Tyr Arg Asn Asn Ile Asp Asp Val Val Arg Phe Leu Asp
165 170 175
Ser Lys His Lys Asn His Tyr Lys Ile Tyr Asn Leu Cys Ala Glu Arg
180 185 190
His Tyr Asp Thr Ala Lys Phe Asn Cys Arg Val Ala Gln Tyr Pro Phe
195 200 205
Glu Asp His Asn Pro Pro Gln Leu Glu Leu Ile Lys Pro Phe Cys Glu
210 215 220
Asp Leu Asp Gln Trp Leu Ser Glu Asp Asp Asn His Val Ala Ala Ile
225 230 235 240
His Cys Lys Ala Gly Lys Gly Arg Thr Gly Val Met Ile Cys Ala Tyr
245 250 255
Leu Leu His Arg Gly Lys Phe Leu Lys Ala Gln Glu Ala Leu Asp Phe
260 265 270
Tyr Gly Glu Val Arg Thr Arg Asp Lys Lys Gly Val Thr Ile Pro Ser
275 280 285
Gln Arg Arg Tyr Val Tyr Tyr Tyr Ser Tyr Leu Leu Lys Asn His Leu
290 295 300
Asp Tyr Arg Pro Val Ala Leu Leu Phe His Lys Met Met Phe Glu Thr
305 310 315 320
Ile Pro Met Phe Ser Gly Gly Thr Cys Asn Pro Gln Phe Val Val Cys
325 330 335
Gln Leu Lys Val Lys Ile Tyr Ser Ser Asn Ser Gly Pro Thr Arg Arg
340 345 350
Glu Asp Lys Phe Met Tyr Phe Glu Phe Pro Gln Pro Leu Pro Val Cys
355 360 365
Gly Asp Ile Lys Val Glu Phe Phe His Lys Gln Asn Lys Met Leu Lys
370 375 380
Lys Asp Lys Met Phe His Phe Trp Val Asn Thr Phe Phe Ile Pro Gly
385 390 395 400
Pro Glu Glu Thr Ser Glu Lys Val Glu Asn Gly Ser Leu Cys Asp Gln
405 410 415
Glu Ile Asp Ser Ile Cys Ser Ile Glu Arg Ala Asp Asn Asp Lys Glu
420 425 430
Tyr Leu Val Leu Thr Leu Thr Lys Asn Asp Leu Asp Lys Ala Asn Lys
435 440 445
Asp Lys Ala Asn Arg Tyr Phe Ser Pro Asn Phe Lys Val Lys Leu Tyr
450 455 460
Phe Thr Lys Thr Val Glu Glu Pro Ser Asn Pro Glu Ala Ser Ser Ser
465 470 475 480
Thr Ser Val Thr Pro Asp Val Ser Asp Asn Glu Pro Asp His Tyr Arg
485 490 495
Tyr Ser Asp Thr Thr Asp Ser Asp Pro Glu Asn Glu Pro Phe Asp Glu
500 505 510
Asp Gln His Thr Gln Ile Thr Lys Val Leu Glu His His His His His
515 520 525
His
<210> 62
<211> 246
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность scFvHER2
<400> 62
Arg Gly Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
1 5 10 15
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
20 25 30
Thr Asp Tyr Thr Met Asp Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
35 40 45
Glu Trp Val Ala Asp Val Asn Pro Asn Ser Gly Gly Ser Ile Tyr Asn
50 55 60
Gln Arg Phe Lys Gly Arg Phe Thr Leu Ser Val Asp Arg Ser Lys Asn
65 70 75 80
Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
85 90 95
Tyr Tyr Cys Ala Arg Asn Leu Gly Pro Ser Phe Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Lys Ala Ser Gln Asp Val Ser Ile Gly Val Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ser Ala Ser Tyr Arg Tyr
180 185 190
Thr Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr
210 215 220
Cys Gln Gln Tyr Tyr Ile Tyr Pro Tyr Thr Phe Gly Gln Gly Thr Lys
225 230 235 240
Val Glu Ile Lys Arg Thr
245
<210> 63
<211> 374
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность UB-scFvHER2-TOM7
<400> 63
Met Gly Ser Ser His His His His His His Ser Ser Gly Leu Val Pro
1 5 10 15
Arg Gly Ser His Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr
20 25 30
Ile Thr Leu Glu Val Glu Pro Ser Asp Thr Ile Glu Asn Val Lys Ala
35 40 45
Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile
50 55 60
Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu Ser Asp Tyr Asn
65 70 75 80
Ile Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly
85 90 95
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
100 105 110
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
115 120 125
Thr Met Asp Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
130 135 140
Ala Asp Val Asn Pro Asn Ser Gly Gly Ser Ile Tyr Asn Gln Arg Phe
145 150 155 160
Lys Gly Arg Phe Thr Leu Ser Val Asp Arg Ser Lys Asn Thr Leu Tyr
165 170 175
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
180 185 190
Ala Arg Asn Leu Gly Pro Ser Phe Tyr Phe Asp Tyr Trp Gly Gln Gly
195 200 205
Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
210 215 220
Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
225 230 235 240
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Lys Ala Ser
245 250 255
Gln Asp Val Ser Ile Gly Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys
260 265 270
Ala Pro Lys Leu Leu Ile Tyr Ser Ala Ser Tyr Arg Tyr Thr Gly Val
275 280 285
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
290 295 300
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
305 310 315 320
Tyr Tyr Ile Tyr Pro Tyr Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
325 330 335
Lys Arg Thr Phe Ala Ile Arg Trp Gly Phe Ile Pro Leu Val Ile Tyr
340 345 350
Leu Gly Phe Lys Arg Gly Ala Asp Pro Gly Met Pro Glu Pro Thr Val
355 360 365
Leu Ser Leu Leu Trp Gly
370
<210> 64
<211> 307
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность scFvHER2-TOM7-myc/His
<400> 64
Met Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
1 5 10 15
Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp
20 25 30
Tyr Thr Met Asp Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
35 40 45
Val Ala Asp Val Asn Pro Asn Ser Gly Gly Ser Ile Tyr Asn Gln Arg
50 55 60
Phe Lys Gly Arg Phe Thr Leu Ser Val Asp Arg Ser Lys Asn Thr Leu
65 70 75 80
Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Asn Leu Gly Pro Ser Phe Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser
130 135 140
Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Lys Ala
145 150 155 160
Ser Gln Asp Val Ser Ile Gly Val Ala Trp Tyr Gln Gln Lys Pro Gly
165 170 175
Lys Ala Pro Lys Leu Leu Ile Tyr Ser Ala Ser Tyr Arg Tyr Thr Gly
180 185 190
Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu
195 200 205
Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln
210 215 220
Gln Tyr Tyr Ile Tyr Pro Tyr Thr Phe Gly Gln Gly Thr Lys Val Glu
225 230 235 240
Ile Lys Arg Thr Phe Ala Ile Arg Trp Gly Phe Ile Pro Leu Val Ile
245 250 255
Tyr Leu Gly Phe Lys Arg Gly Ala Asp Pro Gly Met Pro Glu Pro Thr
260 265 270
Val Leu Ser Leu Leu Trp Gly Leu Glu Ser Arg Gly Pro Phe Glu Gln
275 280 285
Lys Leu Ile Ser Glu Glu Asp Leu Asn Met His Thr Gly His His His
290 295 300
His His His
305
<210> 65
<211> 251
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность scFvMEL
<400> 65
Arg Gly Gly Thr Asp Ile Val Met Thr Gln Ser Gln Lys Phe Met Ser
1 5 10 15
Thr Ser Val Gly Asp Arg Val Ser Val Thr Cys Lys Ala Ser Gln Asn
20 25 30
Val Asp Thr Asn Val Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ser Pro
35 40 45
Glu Pro Leu Leu Phe Ser Ala Ser Tyr Arg Tyr Thr Gly Val Pro Asp
50 55 60
Arg Phe Thr Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Asn Val Gln Ser Glu Asp Leu Ala Glu Tyr Phe Cys Gln Gln Tyr Asn
85 90 95
Ser Tyr Pro Leu Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Gly
100 105 110
Ser Thr Ser Gly Ser Gly Lys Pro Gly Ser Gly Glu Gly Ser Thr Lys
115 120 125
Gly Glu Val Lys Val Glu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Met Lys Leu Ser Cys Val Val Ser Gly Phe Thr Phe Gly Asn
145 150 155 160
Tyr Trp Met Asn Trp Val Arg Gln Ser Pro Glu Lys Gly Leu Glu Trp
165 170 175
Ile Ala Glu Ile Arg Leu Lys Ser Asn Asn Phe Ala Arg Tyr Tyr Ala
180 185 190
Glu Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Ser
195 200 205
Ser Val Tyr Leu Gln Met Ile Asn Leu Arg Ala Glu Asp Thr Gly Ile
210 215 220
Tyr Tyr Cys Thr Ser Tyr Gly Asn Tyr Val Gly His Tyr Phe Asp His
225 230 235 240
Trp Gly Gln Gly Thr Thr Leu Thr Val Ser Ser
245 250
<210> 66
<211> 378
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность UB-scFvMEL-TOM7
<400> 66
Met Gly Ser Ser His His His His His His Ser Ser Gly Leu Val Pro
1 5 10 15
Arg Gly Ser His Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr
20 25 30
Ile Thr Leu Glu Val Glu Pro Ser Asp Thr Ile Glu Asn Val Lys Ala
35 40 45
Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile
50 55 60
Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu Ser Asp Tyr Asn
65 70 75 80
Ile Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly
85 90 95
Thr Asp Ile Met Thr Gln Ser Gln Lys Phe Met Ser Thr Ser Val Gly
100 105 110
Asp Arg Val Ser Val Thr Cys Lys Ala Ser Gln Asn Val Asp Thr Asn
115 120 125
Val Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ser Pro Glu Pro Leu Leu
130 135 140
Phe Ser Ala Ser Tyr Arg Tyr Thr Gly Val Pro Asp Arg Phe Thr Gly
145 150 155 160
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Asn Val Gln Ser
165 170 175
Glu Asp Leu Ala Glu Tyr Phe Cys Gln Gln Tyr Asn Ser Tyr Pro Leu
180 185 190
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Gly Ser Thr Ser Gly
195 200 205
Ser Gly Lys Pro Gly Ser Gly Glu Gly Ser Thr Lys Gly Glu Val Lys
210 215 220
Val Glu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Met Lys
225 230 235 240
Leu Ser Cys Val Val Ser Gly Phe Thr Phe Gly Asn Tyr Trp Met Asn
245 250 255
Trp Val Arg Gln Ser Pro Glu Lys Gly Leu Glu Trp Ile Ala Glu Ile
260 265 270
Arg Leu Lys Ser Asn Asn Phe Ala Arg Tyr Tyr Ala Glu Ser Val Lys
275 280 285
Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Ser Ser Val Tyr Leu
290 295 300
Gln Met Ile Asn Leu Arg Ala Glu Asp Thr Gly Ile Tyr Tyr Cys Thr
305 310 315 320
Ser Tyr Gly Asn Tyr Val Gly His Tyr Phe Asp His Trp Gly Gln Gly
325 330 335
Thr Thr Leu Thr Val Ser Ser Phe Ala Ile Arg Trp Gly Phe Ile Pro
340 345 350
Leu Val Ile Tyr Leu Gly Phe Lys Arg Gly Ala Asp Pro Gly Met Pro
355 360 365
Glu Pro Thr Val Leu Ser Leu Leu Trp Gly
370 375
<210> 67
<211> 312
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность scFvMEL-TOM7-myc/His
<400> 67
Met Thr Asp Ile Val Met Thr Gln Ser Gln Lys Phe Met Ser Thr Ser
1 5 10 15
Val Gly Asp Arg Val Ser Val Thr Cys Lys Ala Ser Gln Asn Val Asp
20 25 30
Thr Asn Val Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ser Pro Glu Pro
35 40 45
Leu Leu Phe Ser Ala Ser Tyr Arg Tyr Thr Gly Val Pro Asp Arg Phe
50 55 60
Thr Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Asn Val
65 70 75 80
Gln Ser Glu Asp Leu Ala Glu Tyr Phe Cys Gln Gln Tyr Asn Ser Tyr
85 90 95
Pro Leu Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Gly Ser Thr
100 105 110
Ser Gly Ser Gly Lys Pro Gly Ser Gly Glu Gly Ser Thr Lys Gly Glu
115 120 125
Val Lys Val Glu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
130 135 140
Met Lys Leu Ser Cys Val Val Ser Gly Phe Thr Phe Gly Asn Tyr Trp
145 150 155 160
Met Asn Trp Val Arg Gln Ser Pro Glu Lys Gly Leu Glu Trp Ile Ala
165 170 175
Glu Ile Arg Leu Lys Ser Asn Asn Phe Ala Arg Tyr Tyr Ala Glu Ser
180 185 190
Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Ser Ser Val
195 200 205
Tyr Leu Gln Met Ile Asn Leu Arg Ala Glu Asp Thr Gly Ile Tyr Tyr
210 215 220
Cys Thr Ser Tyr Gly Asn Tyr Val Gly His Tyr Phe Asp His Trp Gly
225 230 235 240
Gln Gly Thr Thr Leu Thr Val Ser Ser Phe Ala Ile Arg Trp Gly Phe
245 250 255
Ile Pro Leu Val Ile Tyr Leu Gly Phe Lys Arg Gly Ala Asp Pro Gly
260 265 270
Met Pro Glu Pro Thr Val Leu Ser Leu Leu Trp Gly Leu Glu Ser Arg
275 280 285
Gly Pro Phe Glu Gln Lys Leu Ile Ser Glu Glu Asp Leu Asn Met His
290 295 300
Thr Gly His His His His His His
305 310
<210> 68
<211> 250
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность scFvPD-L1
<400> 68
Met Asp Ile Val Met Ser Gln Ser Pro Ser Ser Leu Ala Val Ser Ala
1 5 10 15
Gly Glu Lys Val Thr Met Ser Cys Lys Ser Ser Gln Ser Leu Leu Asn
20 25 30
Ser Arg Thr Arg Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly
35 40 45
Gln Ser Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly
50 55 60
Val Pro Asp Arg Phe Thr Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu
65 70 75 80
Thr Ile Ser Ser Val Gln Ala Glu Asp Leu Ala Val Tyr Tyr Cys Gln
85 90 95
Gln Ser Tyr Asp Val Val Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu
100 105 110
Lys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
115 120 125
Gln Val Gln Val Gln Gln Ser Gly Ala Glu Leu Ala Glu Pro Gly Ala
130 135 140
Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr Ile Phe Thr Ser Tyr
145 150 155 160
Trp Met His Trp Leu Lys Gln Arg Pro Gly Gln Gly Leu Glu Trp Ile
165 170 175
Gly Tyr Ile Asn Pro Ser Ser Asp Tyr Asn Glu Tyr Ser Glu Lys Phe
180 185 190
Met Asp Lys Ala Thr Leu Thr Ala Asp Lys Ala Ser Thr Thr Ala Tyr
195 200 205
Met Gln Leu Ile Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Ser Gly Trp Leu Val His Gly Asp Tyr Tyr Phe Asp Tyr Trp
225 230 235 240
Gly Gln Gly Thr Thr Leu Thr Val Ser Ser
245 250
<210> 69
<211> 315
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность scFvPD-L1-TOM7-myc/His
<400> 69
Met Asp Ile Val Met Ser Gln Ser Pro Ser Ser Leu Ala Val Ser Ala
1 5 10 15
Gly Glu Lys Val Thr Met Ser Cys Lys Ser Ser Gln Ser Leu Leu Asn
20 25 30
Ser Arg Thr Arg Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly
35 40 45
Gln Ser Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly
50 55 60
Val Pro Asp Arg Phe Thr Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu
65 70 75 80
Thr Ile Ser Ser Val Gln Ala Glu Asp Leu Ala Val Tyr Tyr Cys Gln
85 90 95
Gln Ser Tyr Asp Val Val Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu
100 105 110
Lys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
115 120 125
Gln Val Gln Val Gln Gln Ser Gly Ala Glu Leu Ala Glu Pro Gly Ala
130 135 140
Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr Ile Phe Thr Ser Tyr
145 150 155 160
Trp Met His Trp Leu Lys Gln Arg Pro Gly Gln Gly Leu Glu Trp Ile
165 170 175
Gly Tyr Ile Asn Pro Ser Ser Asp Tyr Asn Glu Tyr Ser Glu Lys Phe
180 185 190
Met Asp Lys Ala Thr Leu Thr Ala Asp Lys Ala Ser Thr Thr Ala Tyr
195 200 205
Met Gln Leu Ile Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Ser Gly Trp Leu Val His Gly Asp Tyr Tyr Phe Asp Tyr Trp
225 230 235 240
Gly Gln Gly Thr Thr Leu Thr Val Ser Ser Leu Glu Phe Ala Ile Arg
245 250 255
Trp Gly Phe Ile Pro Leu Val Ile Tyr Leu Gly Phe Lys Arg Gly Ala
260 265 270
Asp Pro Gly Met Pro Glu Pro Thr Val Leu Ser Leu Leu Trp Gly Leu
275 280 285
Glu Ser Arg Gly Pro Phe Glu Gln Lys Leu Ile Ser Glu Glu Asp Leu
290 295 300
Asn Met His Thr Gly His His His His His His
305 310 315
<210> 70
<211> 15
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Линкер
<400> 70
ggggsggggs ggggs
15
<210> 71
<211> 75
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность убиквитина
<400> 71
Gln Leu Phe Val Lys Thr Leu Thr Gly Lys Thr Val Thr Leu Glu Val
1 5 10 15
Glu Ser Ser Asp Thr Ile Asp Asn Val Lys Ser Lys Ile Gln Asp Lys
20 25 30
Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys Gln
35 40 45
Leu Glu Asp Gly Arg Thr Leu Ser Asp Tyr Asn Ile Gln Lys Glu Ser
50 55 60
Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly
65 70 75
<210> 72
<211> 225
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность, кодирующая убиквитин
<400> 72
caacttttcg tcaaaactct aacagggaag actgtaaccc tagaggttga atcttccgac
60
actattgaca acgtcaaaag taaaattcaa gataaagaag gtatccctcc ggatcagcag
120
agattgattt ttgctggtaa gcaactagaa gatggtagaa ccttgtctga ctacaacatc
180
caaaaggaat ctactcttca cttggtgttg agactccgcg gtggt
225
<210> 73
<211> 400
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность p53
<400> 73
Glu Glu Pro Gln Ser Asp Pro Ser Val Glu Pro Pro Leu Ser Gln Glu
1 5 10 15
Thr Phe Ser Asp Leu Trp Lys Leu Leu Pro Glu Asn Asn Val Leu Ser
20 25 30
Pro Leu Pro Ser Gln Ala Met Asp Asp Leu Met Leu Ser Pro Asp Asp
35 40 45
Ile Glu Gln Trp Phe Thr Glu Asp Pro Gly Pro Asp Glu Ala Pro Arg
50 55 60
Met Pro Glu Ala Ala Pro Arg Val Ala Pro Ala Pro Ala Ala Pro Thr
65 70 75 80
Pro Ala Ala Pro Ala Pro Ala Pro Ser Trp Pro Leu Ser Ser Ser Val
85 90 95
Pro Ser Gln Lys Thr Tyr Gln Gly Ser Tyr Gly Phe Arg Leu Gly Phe
100 105 110
Leu His Ser Gly Thr Ala Lys Ser Val Thr Cys Thr Tyr Ser Pro Ala
115 120 125
Leu Asn Lys Met Phe Cys Gln Leu Ala Lys Thr Cys Pro Val Gln Leu
130 135 140
Trp Val Asp Ser Thr Pro Pro Pro Gly Thr Arg Val Arg Ala Met Ala
145 150 155 160
Ile Tyr Lys Gln Ser Gln His Met Thr Glu Val Val Arg Arg Cys Pro
165 170 175
His His Glu Arg Cys Ser Asp Ser Asp Gly Leu Ala Pro Pro Gln His
180 185 190
Leu Ile Arg Val Glu Gly Asn Leu Arg Val Glu Tyr Leu Asp Asp Arg
195 200 205
Asn Thr Phe Arg His Ser Val Val Val Pro Tyr Glu Pro Pro Glu Val
210 215 220
Gly Ser Asp Cys Thr Thr Ile His Tyr Asn Tyr Met Cys Asn Ser Ser
225 230 235 240
Cys Met Gly Gly Met Asn Arg Arg Pro Ile Leu Thr Ile Ile Thr Leu
245 250 255
Glu Asp Ser Ser Gly Asn Leu Leu Gly Arg Asn Ser Phe Glu Val Arg
260 265 270
Val Cys Ala Cys Pro Gly Arg Asp Arg Arg Thr Glu Glu Glu Asn Leu
275 280 285
Arg Lys Lys Gly Glu Pro His His Glu Leu Pro Pro Gly Ser Thr Lys
290 295 300
Arg Ala Leu Pro Asn Asn Thr Ser Ser Ser Pro Gln Pro Lys Lys Lys
305 310 315 320
Pro Leu Asp Gly Glu Tyr Phe Thr Leu Gln Ile Arg Gly Arg Glu Arg
325 330 335
Phe Glu Met Phe Arg Glu Leu Asn Glu Ala Leu Glu Leu Lys Asp Ala
340 345 350
Gln Ala Gly Lys Glu Pro Gly Gly Ser Arg Ala His Ser Ser His Leu
355 360 365
Lys Ser Lys Lys Gly Gln Ser Thr Ser Arg His Lys Lys Leu Met Phe
370 375 380
Lys Thr Glu Gly Pro Asp Ser Asp Leu Glu His His His His His His
385 390 395 400
<210> 74
<211> 1203
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность, кодирующая p53
<400> 74
gaggagccgc agtcagatcc tagcgtcgag ccccctctga gtcaggaaac attttcagac
60
ctgtggaaac tacttcctga aaacaacgtt ctgtccccct tgccgtccca agcaatggat
120
gatttgatgc tgtccccgga cgatattgaa caatggttca ctgaagaccc aggtccagat
180
gaagctccca gaatgccaga ggctgctccc cgcgtggccc ctgcaccagc agctcctaca
240
ccggcggccc ctgcaccagc cccctcctgg cccctgtcat cttctgtccc ttcccagaaa
300
acctaccagg gcagctacgg tttccgtctg ggcttcttgc attctgggac agccaagtct
360
gtgacttgca cgtactcccc tgccctcaac aagatgtttt gccaactggc caagacctgc
420
cctgtgcagc tgtgggttga ttccacaccc ccgcccggca cccgcgtccg cgccatggcc
480
atctacaagc agtcacagca catgacggag gttgtgaggc gctgccccca ccatgagcgc
540
tgctcagata gcgatggtct ggcccctcct cagcatctta tccgagtgga aggaaatttg
600
cgtgtggagt atttggatga cagaaacact tttcgacata gtgtggtggt gccctatgag
660
ccgcctgagg ttggctctga ctgtaccacc atccactaca actacatgtg taacagttcc
720
tgcatgggcg gcatgaaccg gaggcccatc ctcaccatca tcacactgga agactccagt
780
ggtaatctac tgggacggaa cagctttgag gtgcgtgttt gtgcctgtcc tgggagagac
840
cggcgcacag aggaagagaa tctccgcaag aaaggggagc ctcaccacga gctgccccca
900
gggagcacta agcgagcact gcccaacaac accagctcct ctccccagcc aaagaagaaa
960
ccactggatg gagaatattt cacccttcag atccgtgggc gtgagcgctt cgagatgttc
1020
cgagagctga atgaggcctt ggaactcaag gatgcccagg ctgggaagga gccagggggg
1080
agcagggctc actccagcca cctgaagtcc aaaaagggtc agtctacctc ccgccataaa
1140
aaactcatgt tcaagacaga agggcctgac tcagacctcg agcaccacca ccaccaccac
1200
tag
1203
<210> 75
<211> 29
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> TOM70 S. cerevisiae
<400> 75
Met Lys Ser Phe Ile Thr Arg Asn Lys Thr Ala Ile Phe Ala Thr Val
1 5 10 15
Ala Ala Thr Gly Thr Ala Ile Gly Ala Tyr Tyr Tyr Tyr
20 25
<210> 76
<211> 60
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> TOM70 Homo sapiens
<400> 76
Met Ala Ala Ser Lys Pro Val Glu Ala Ala Val Val Ala Ala Ala Val
1 5 10 15
Pro Ser Ser Gly Ser Gly Val Gly Gly Gly Gly Thr Ala Gly Pro Gly
20 25 30
Thr Gly Gly Leu Pro Arg Trp Gln Leu Ala Leu Ala Val Gly Ala Pro
35 40 45
Leu Leu Leu Gly Ala Gly Ala Ile Tyr Leu Trp Ser
50 55 60
<210> 77
<211> 29
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> TOM20 S. cerevisiae
<400> 77
Met Ser Gln Ser Asn Pro Ile Leu Arg Gly Leu Ala Ile Thr Thr Ala
1 5 10 15
Ile Ala Ala Leu Ser Ala Thr Gly Tyr Ala Ile Tyr Phe
20 25
<210> 78
<211> 24
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> TOM20 Homo sapiens
<400> 78
Met Val Gly Arg Asn Ser Ala Ile Ala Ala Gly Val Cys Gly Ala Leu
1 5 10 15
Phe Ile Gly Tyr Cys Ile Tyr Phe
20
<210> 79
<211> 22
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> OM45 S. cerevisiae
<400> 79
Met Ser Ser Arg Ile Ile Val Gly Ser Ala Ala Leu Ala Ala Ala Ile
1 5 10 15
Thr Ala Ser Ile Met Val
20
<210> 80
<211> 23
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> TOM5 S. cerevisiae
<400> 80
Ala Ala Tyr Val Ala Ala Phe Leu Trp Val Ser Pro Met Ile Trp His
1 5 10 15
Leu Val Lys Lys Gln Trp Lys
20
<210> 81
<211> 24
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> TOM5 Homo sapiens
<400> 81
Ile Arg Asn Phe Leu Ile Tyr Val Ala Leu Leu Arg Val Thr Pro Phe
1 5 10 15
Ile Leu Lys Lys Leu Asp Ser Ile
20
<210> 82
<211> 41
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> TOM7 S. cerevisiae
<400> 82
Ile Leu Thr Leu Thr His Asn Val Ala His Tyr Gly Trp Ile Pro Phe
1 5 10 15
Val Leu Tyr Leu Gly Trp Ala His Thr Ser Asn Arg Pro Asn Phe Leu
20 25 30
Asn Leu Leu Ser Pro Leu Pro Ser Val
35 40
<210> 83
<211> 35
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> TOM7 Homo sapiens
<400> 83
Phe Ala Ile Arg Trp Gly Phe Ile Pro Leu Val Ile Tyr Leu Gly Phe
1 5 10 15
Lys Arg Gly Ala Asp Pro Gly Met Pro Glu Pro Thr Val Leu Ser Leu
20 25 30
Leu Trp Gly
35
<210> 84
<211> 55
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> TOM22 S. cerevisiae
<400> 84
Leu Ala Trp Thr Leu Thr Thr Thr Ala Leu Leu Leu Gly Val Pro Leu
1 5 10 15
Ser Leu Ser Ile Leu Ala Glu Gln Gln Leu Ile Glu Met Glu Lys Thr
20 25 30
Phe Asp Leu Gln Ser Asp Ala Asn Asn Ile Leu Ala Gln Gly Glu Lys
35 40 45
Asp Ala Ala Ala Thr Ala Asn
50 55
<210> 85
<211> 60
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> TOM22 Homo sapiens
<400> 85
Ala Ala Leu Trp Ile Gly Thr Thr Ser Phe Met Ile Leu Val Leu Pro
1 5 10 15
Val Val Phe Glu Thr Glu Lys Leu Gln Met Glu Gln Gln Gln Gln Leu
20 25 30
Gln Gln Arg Gln Ile Leu Leu Gly Pro Asn Thr Gly Leu Ser Gly Gly
35 40 45
Met Pro Gly Ala Leu Pro Ser Leu Pro Gly Lys Ile
50 55 60
<210> 86
<211> 25
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Fis1 S. cerevisiae
<400> 86
Gly Val Val Val Ala Gly Gly Val Leu Ala Gly Ala Val Ala Val Ala
1 5 10 15
Ser Phe Phe Leu Arg Asn Lys Arg Arg
20 25
<210> 87
<211> 31
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Fis1 Homo sapiens
<400> 87
Gly Leu Val Gly Met Ala Ile Val Gly Gly Met Ala Leu Gly Val Ala
1 5 10 15
Gly Leu Ala Gly Leu Ile Gly Leu Ala Val Ser Lys Ser Lys Ser
20 25 30
<210> 88
<211> 25
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Bcl-2 альфа Homo sapiens
<400> 88
Leu Ser Leu Lys Thr Leu Leu Ser Leu Ala Leu Val Gly Ala Cys Ile
1 5 10 15
Thr Leu Gly Ala Tyr Leu Gly His Lys
20 25
<210> 89
<211> 19
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VAMP1 S. cerevisiae
<400> 89
Met Ile Met Leu Gly Ala Ile Cys Ala Ile Ile Val Val Val Ile Val
1 5 10 15
Arg Arg Asp
<210> 90
<211> 22
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VAMP1 Homo sapiens
<400> 90
Met Met Ile Met Leu Gly Ala Ile Cys Ala Ile Ile Val Val Val Ile
1 5 10 15
Val Ile Tyr Phe Phe Thr
20
<210> 91
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<223> P53-promter-S
<400> 91
gggcatgctc gggcatgccc gggcatgctc gggcatgccc gggcatgctc gggcatgccc
60
60
<210> 92
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<223> P53-promter-AS
<400> 92
gggcatgccc gagcatgccc gggcatgccc gagcatgccc gggcatgccc gagcatgccc
60
60
<---
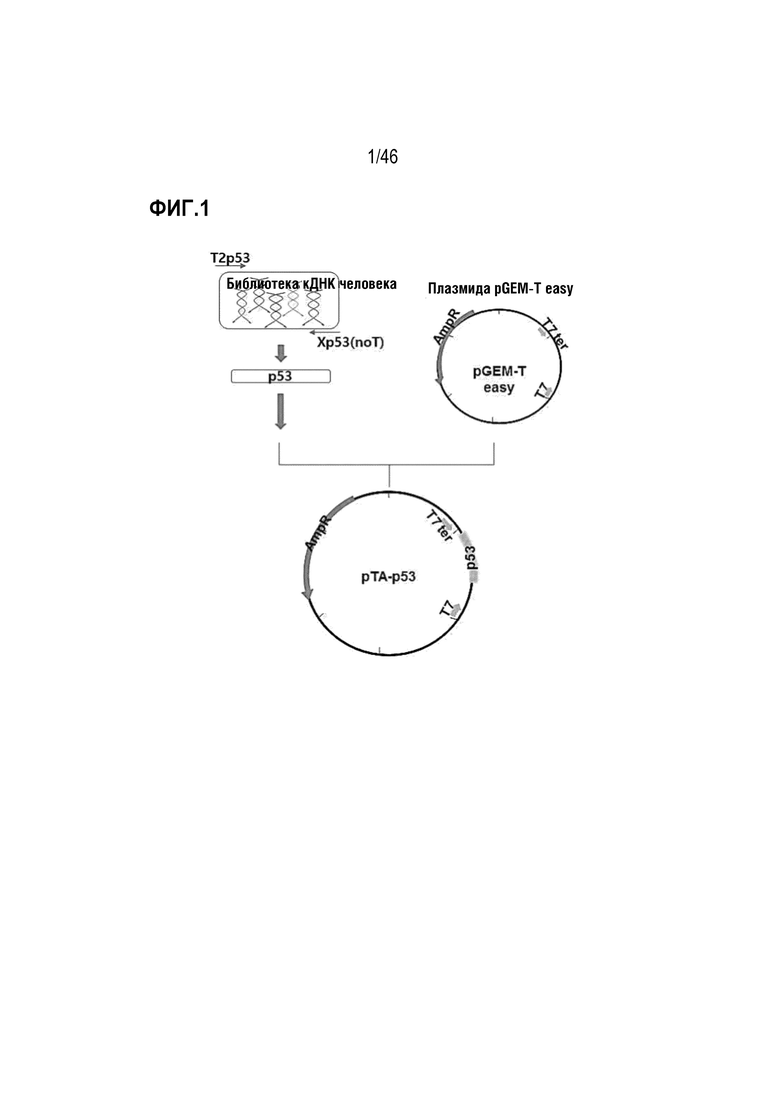
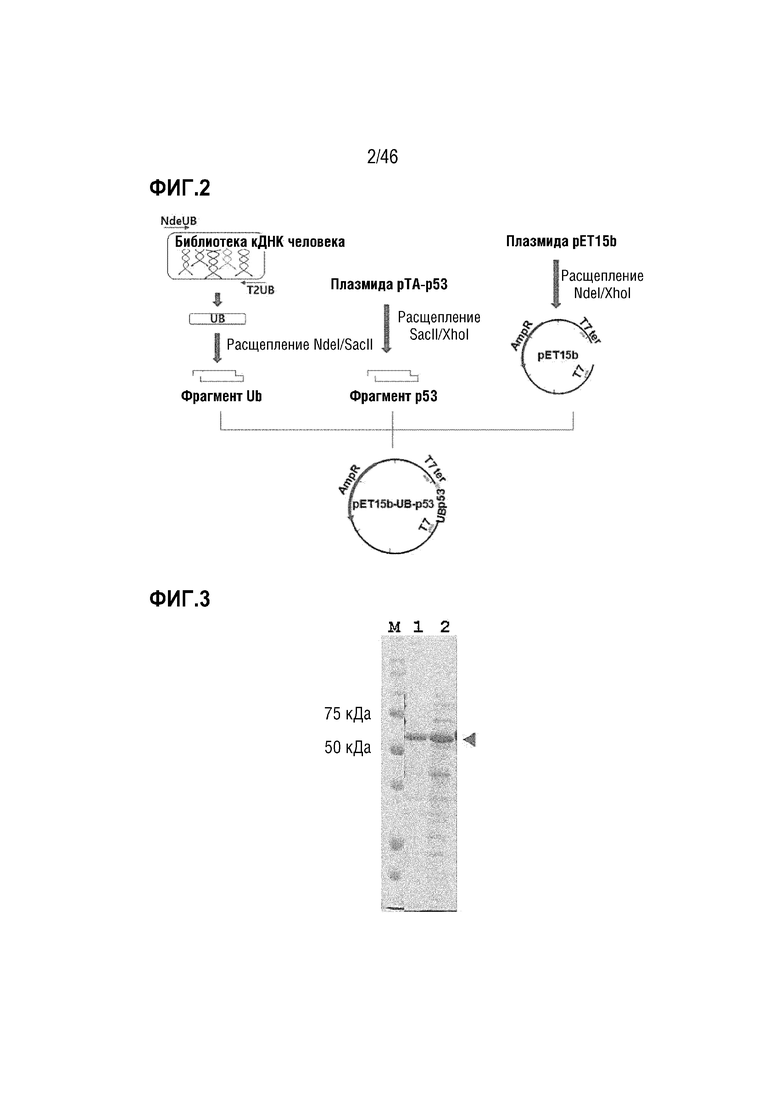
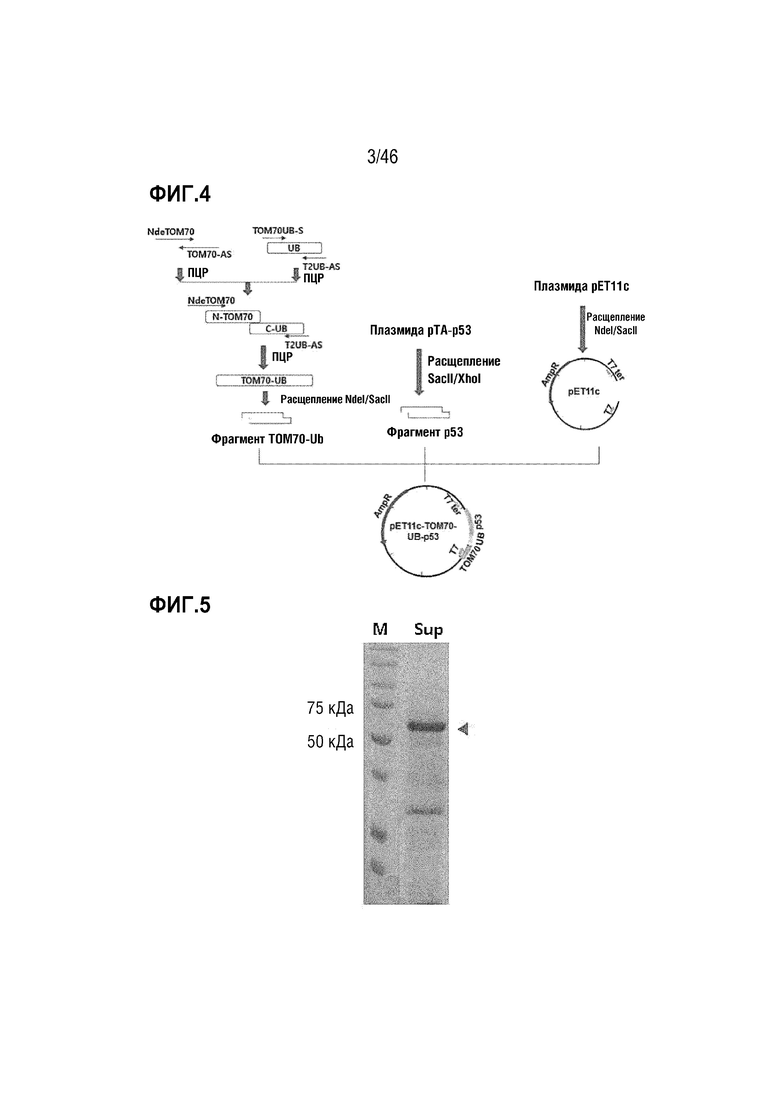
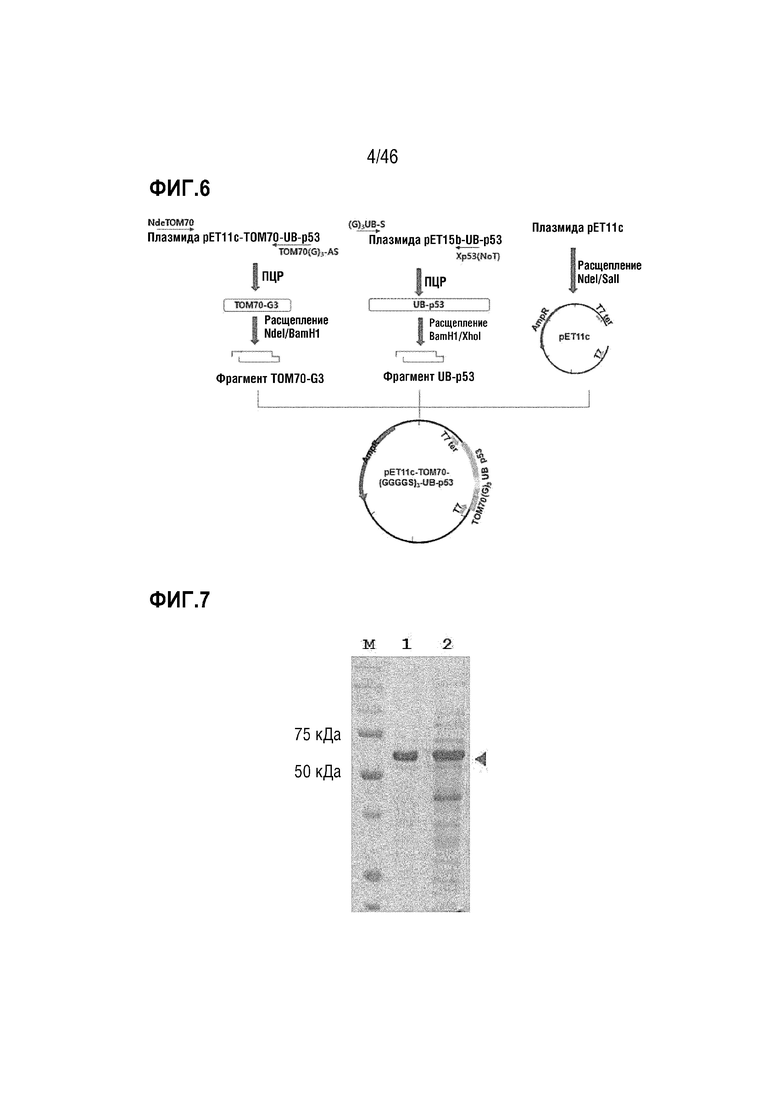
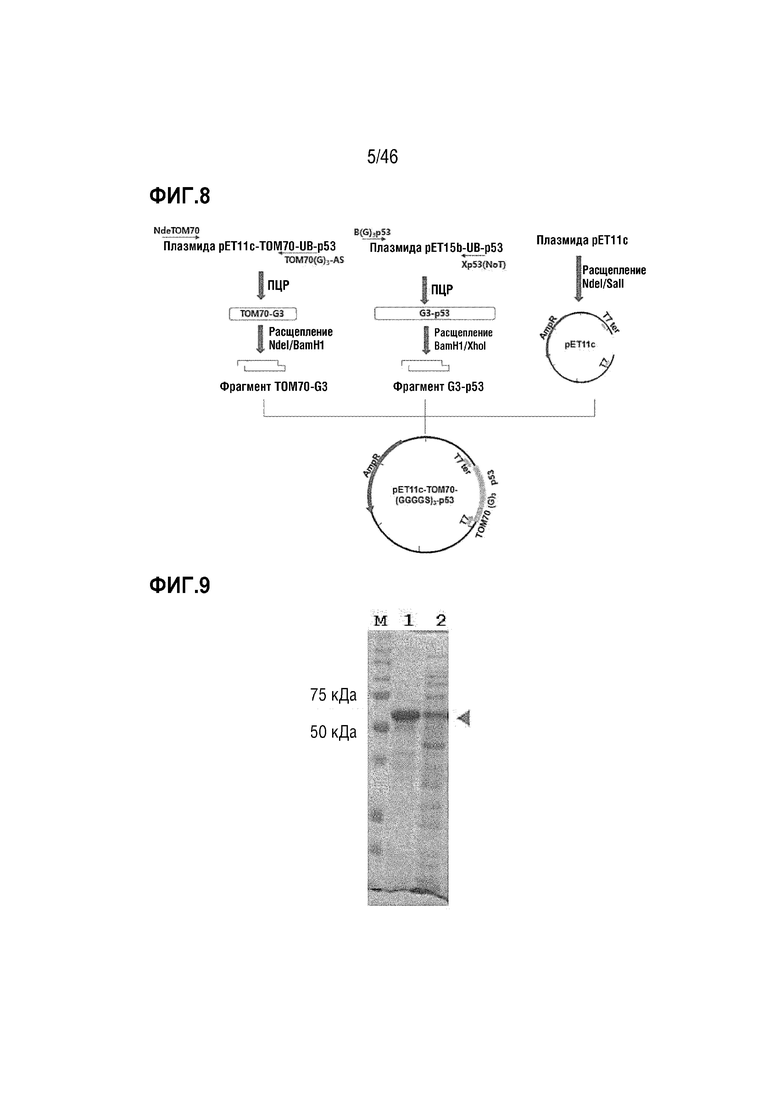
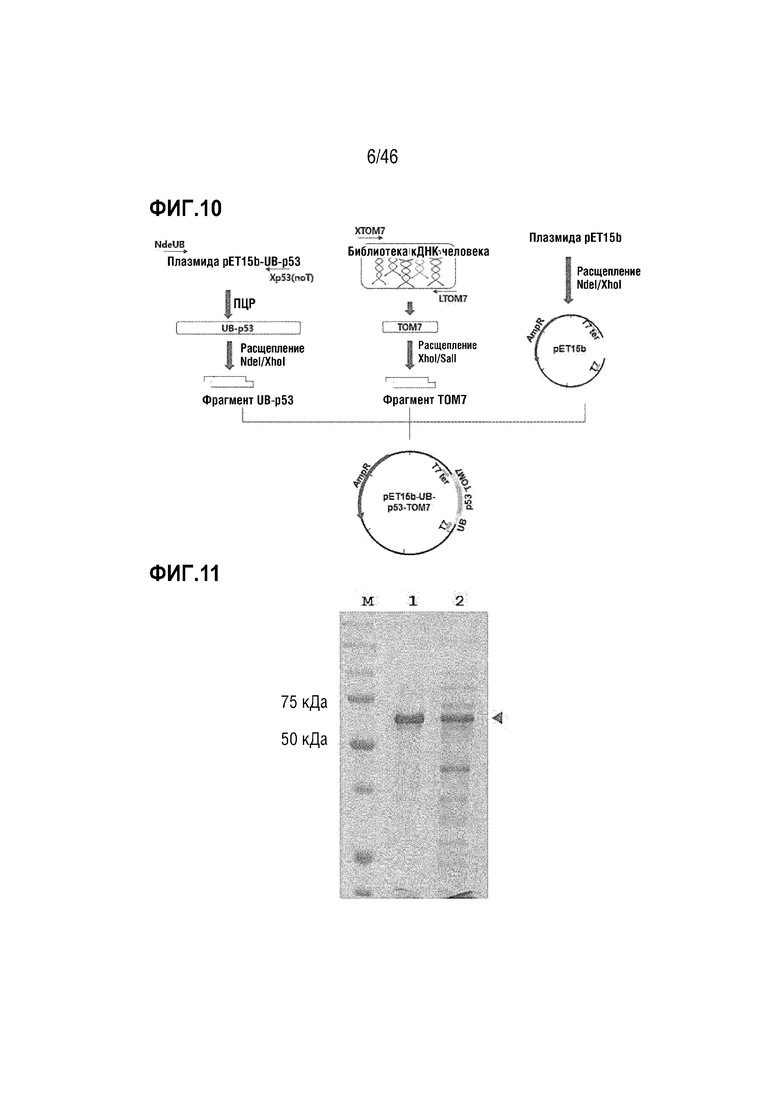
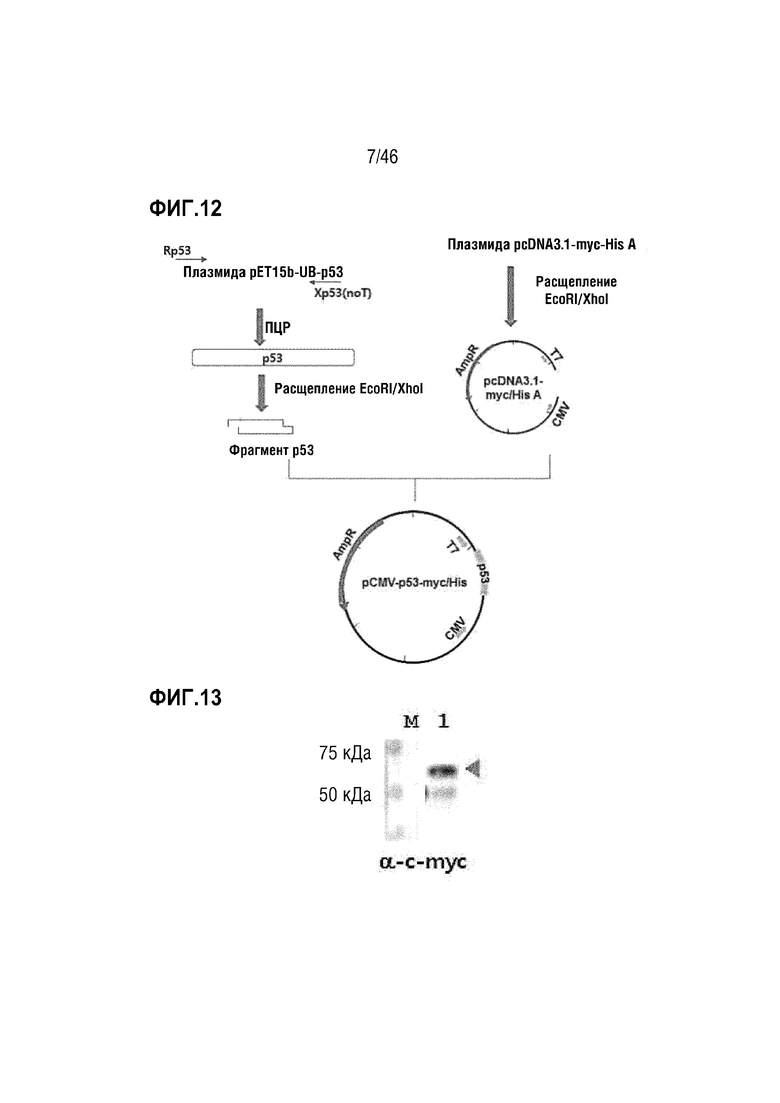
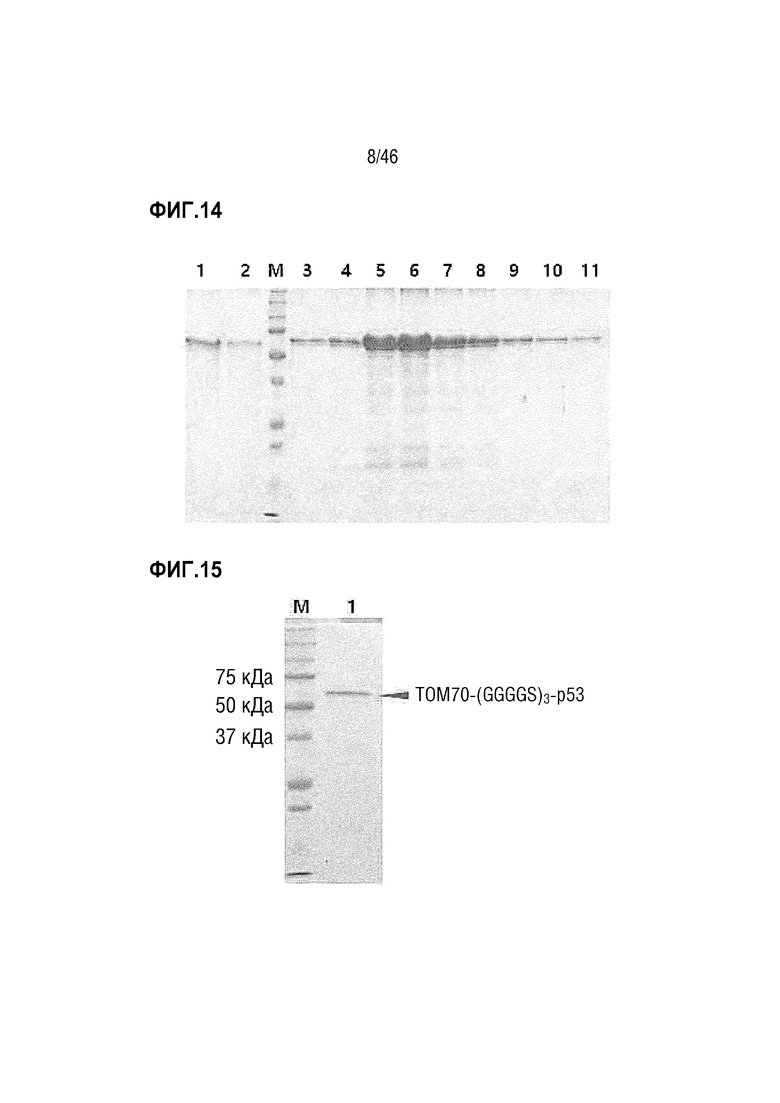

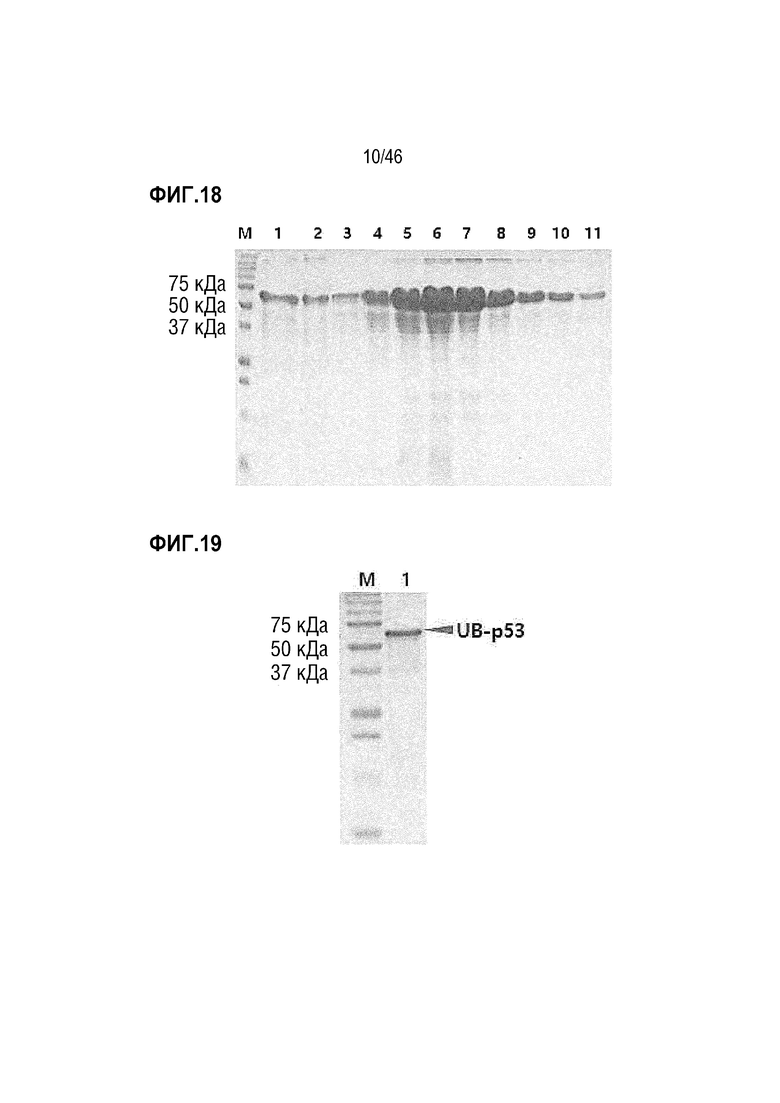
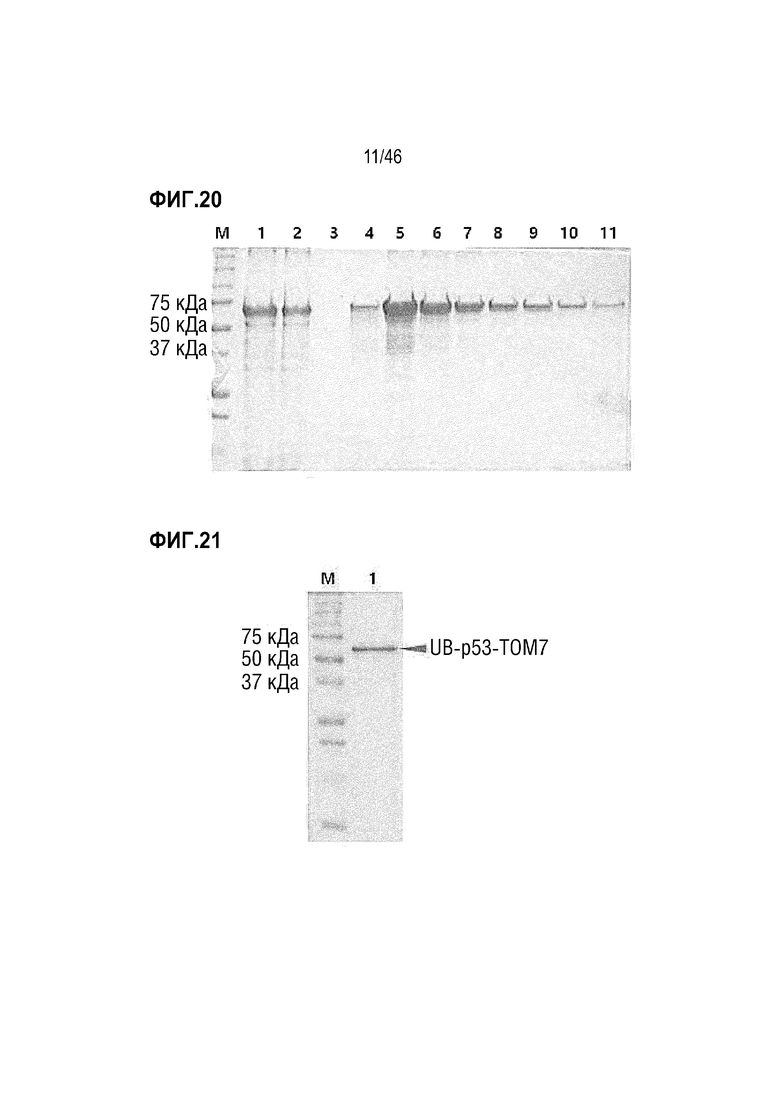
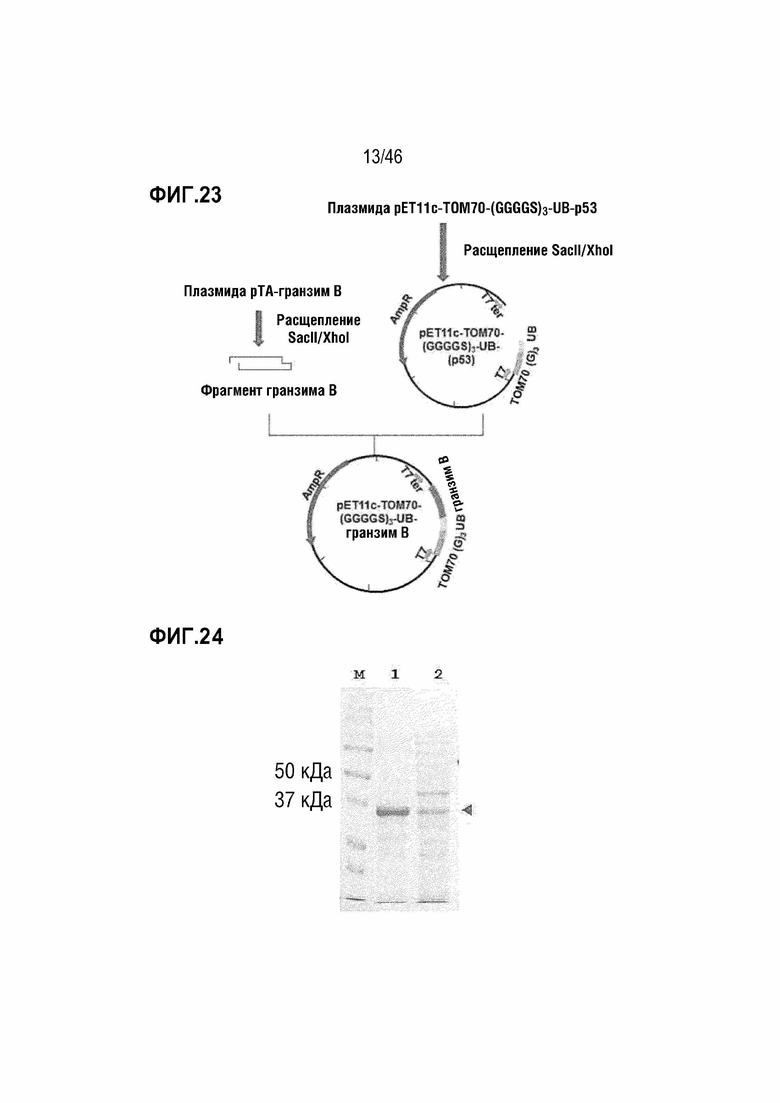
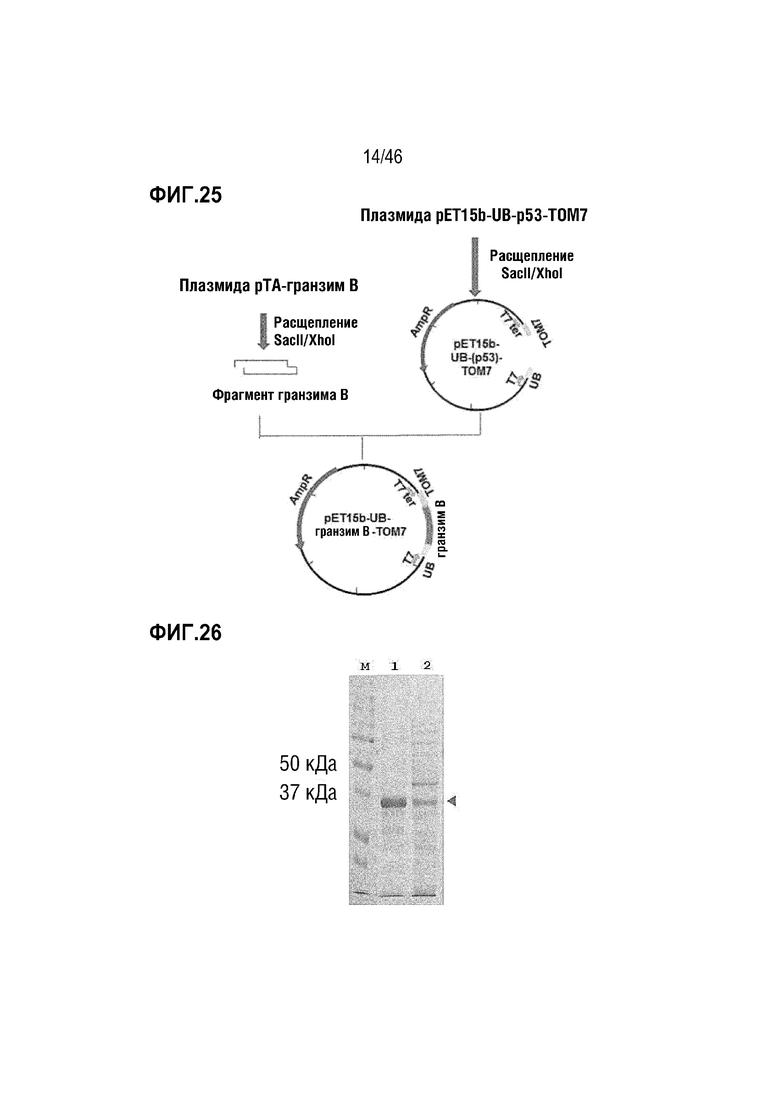
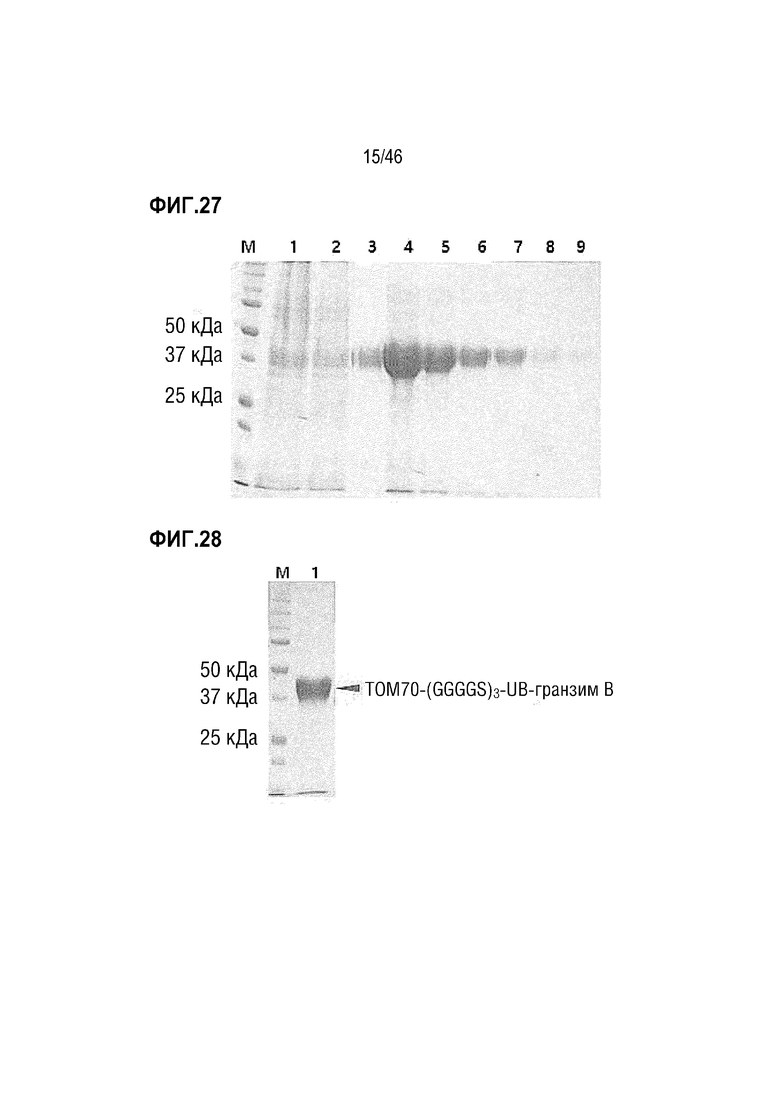
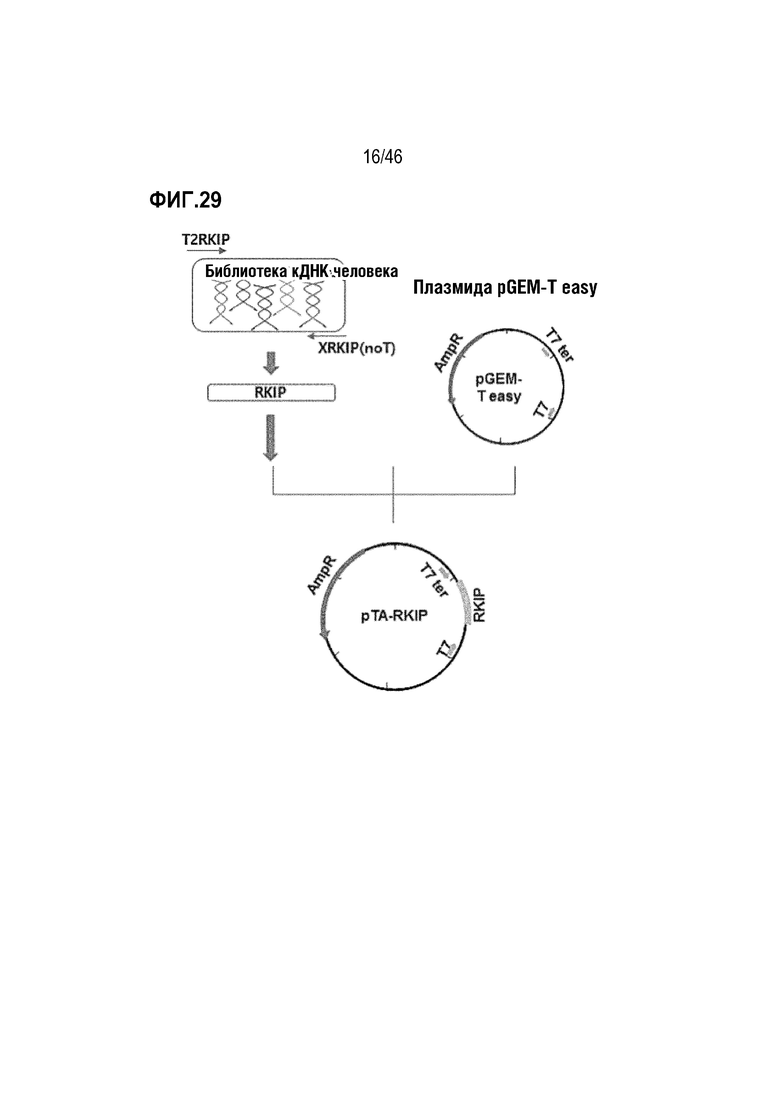

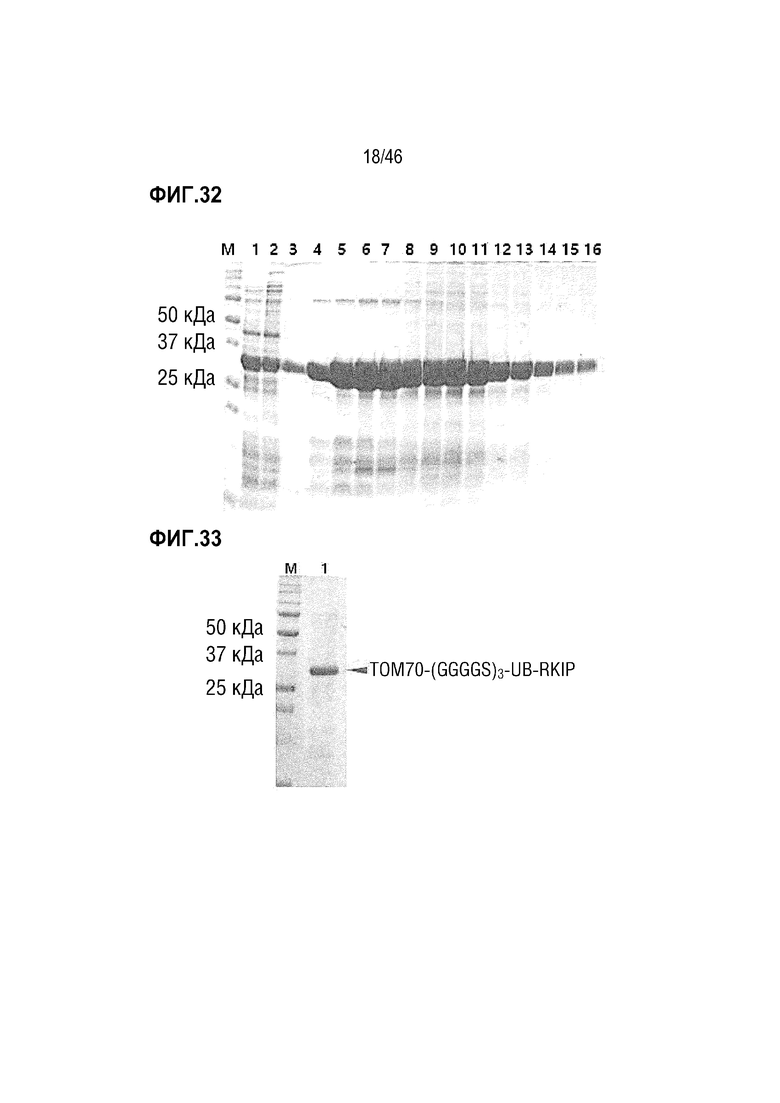
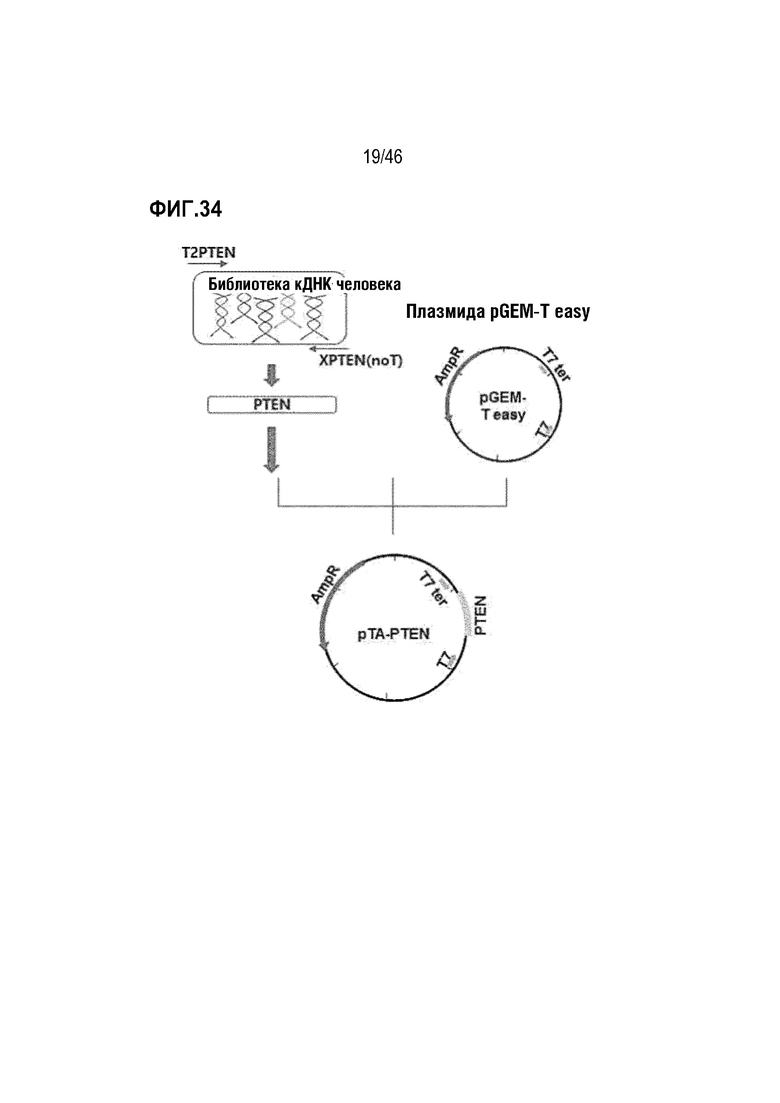

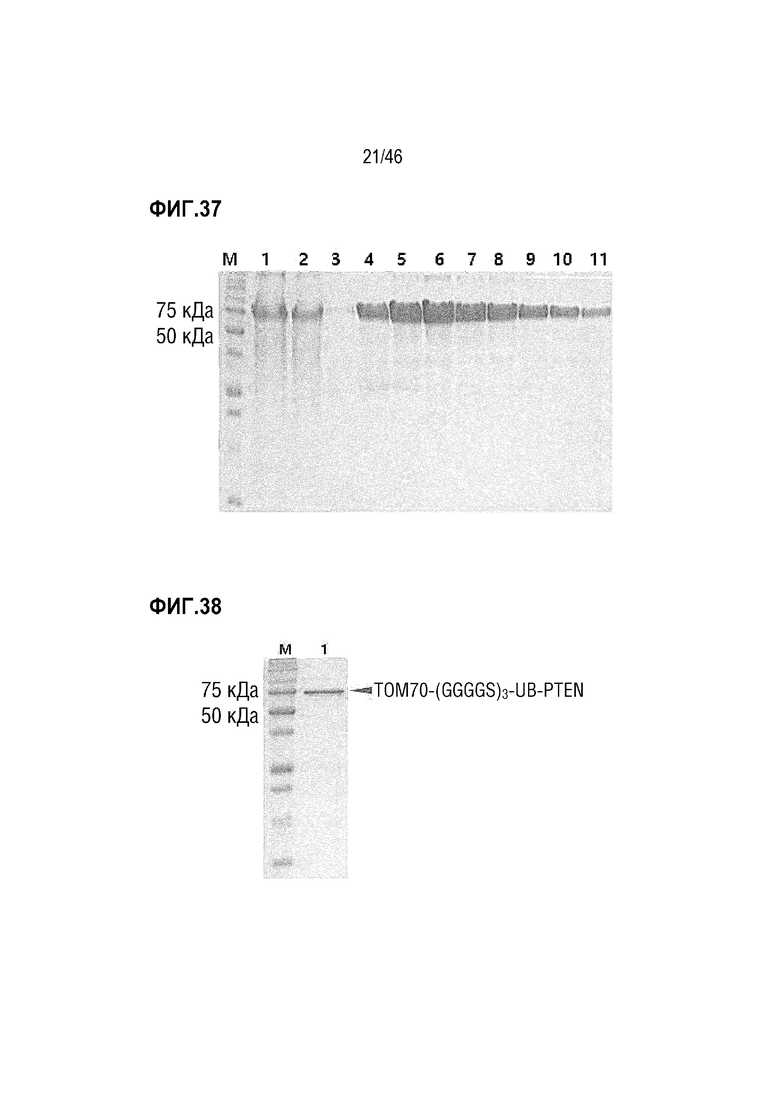




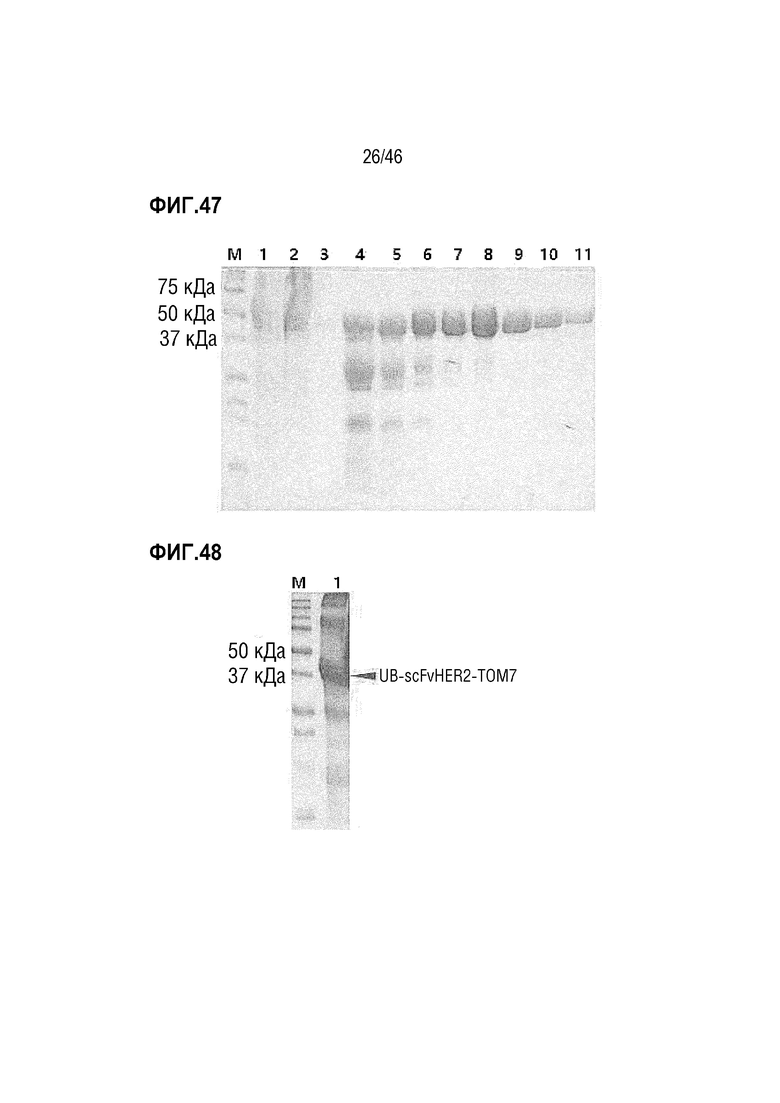
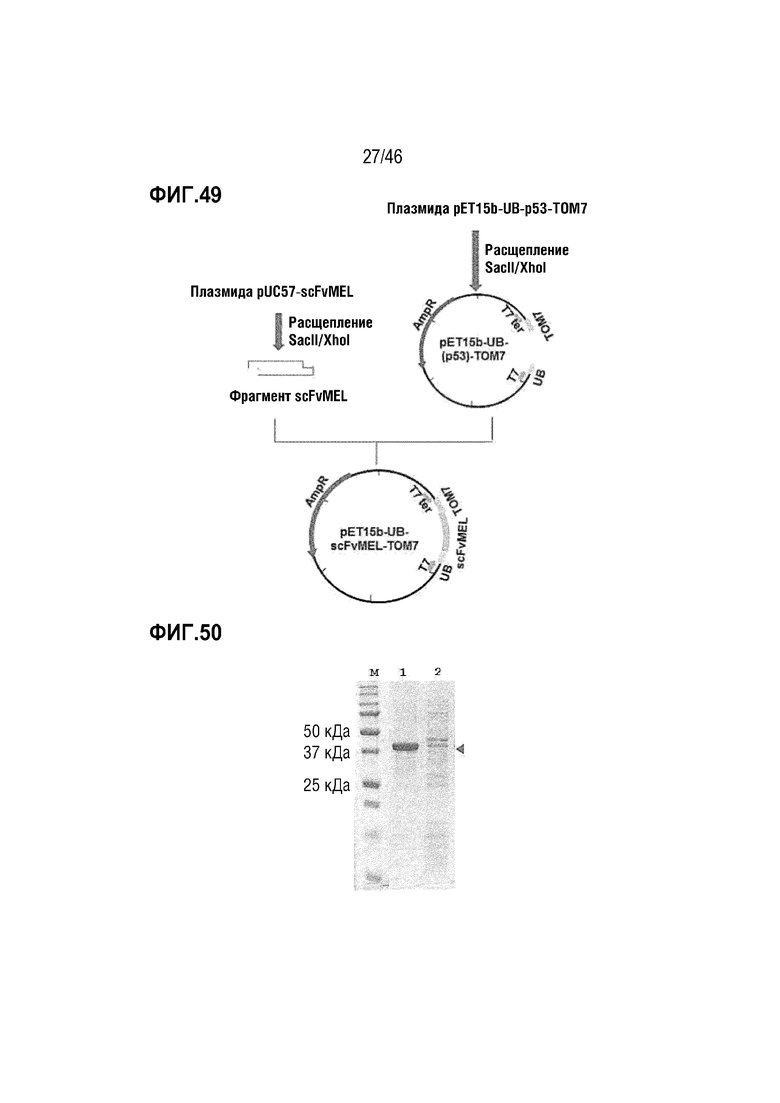
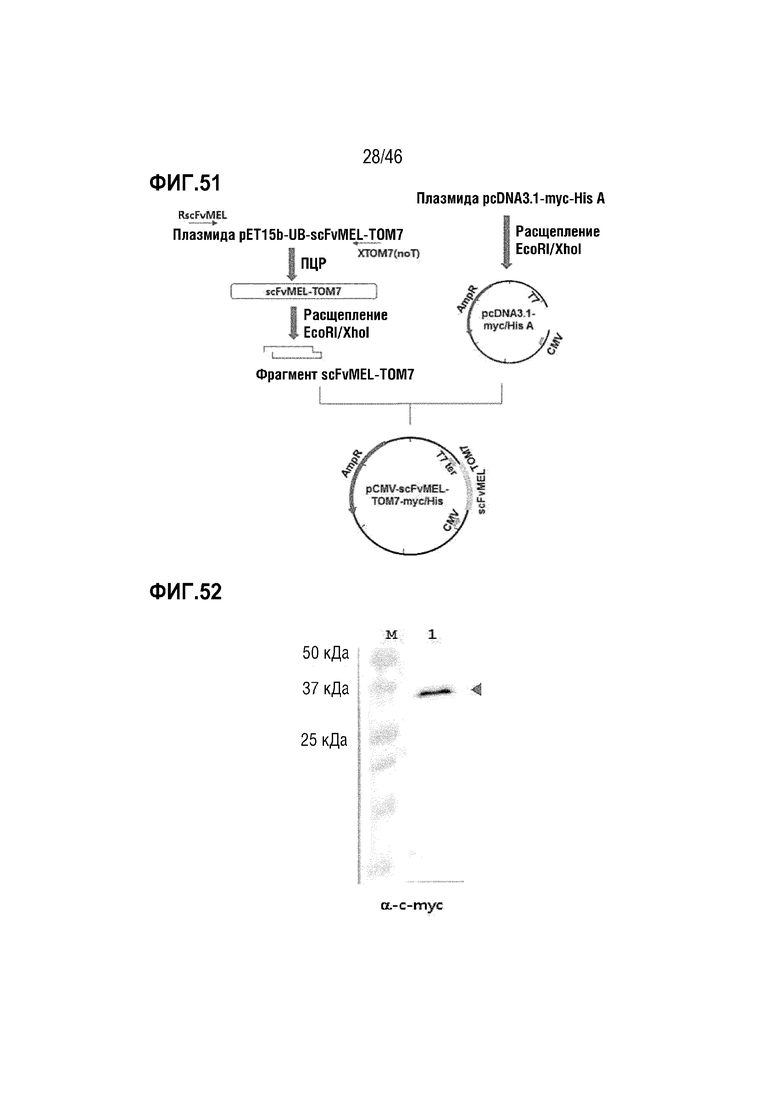
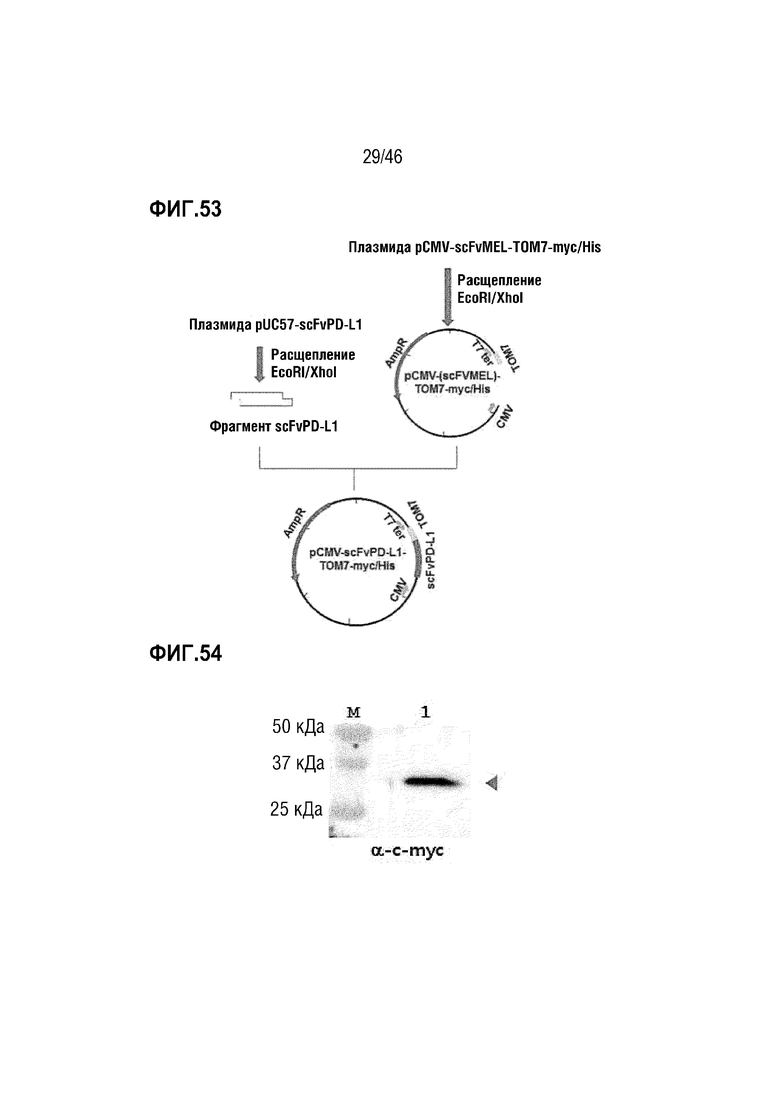
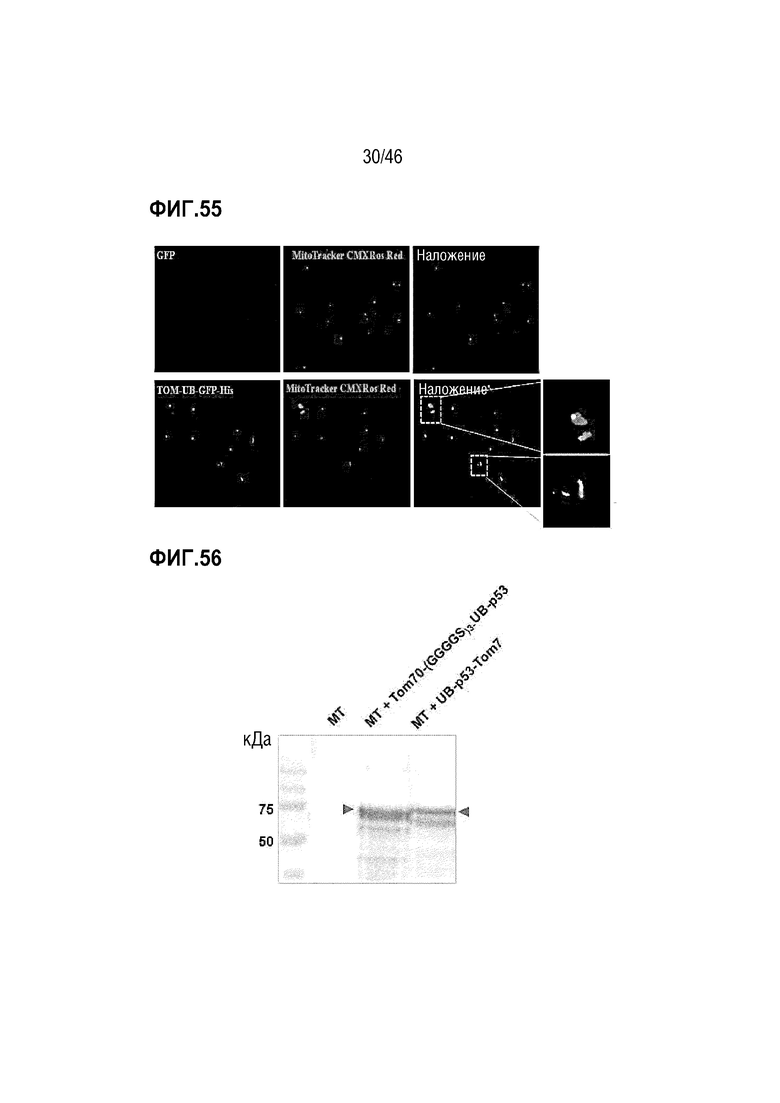
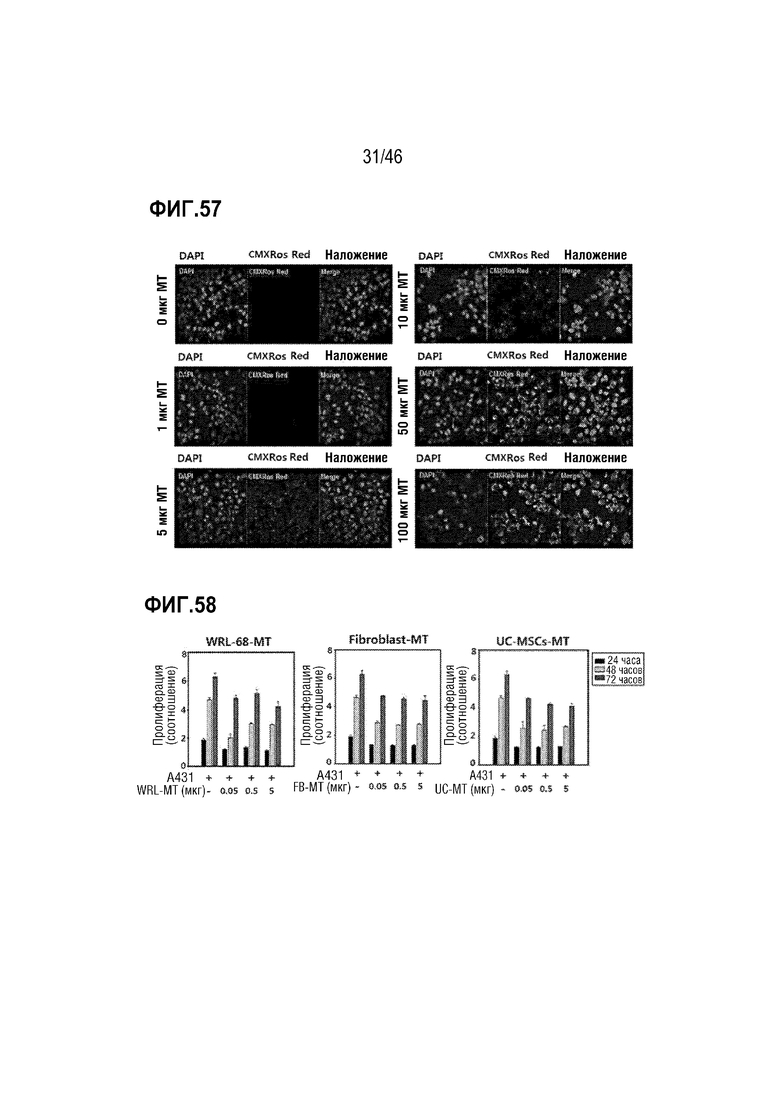
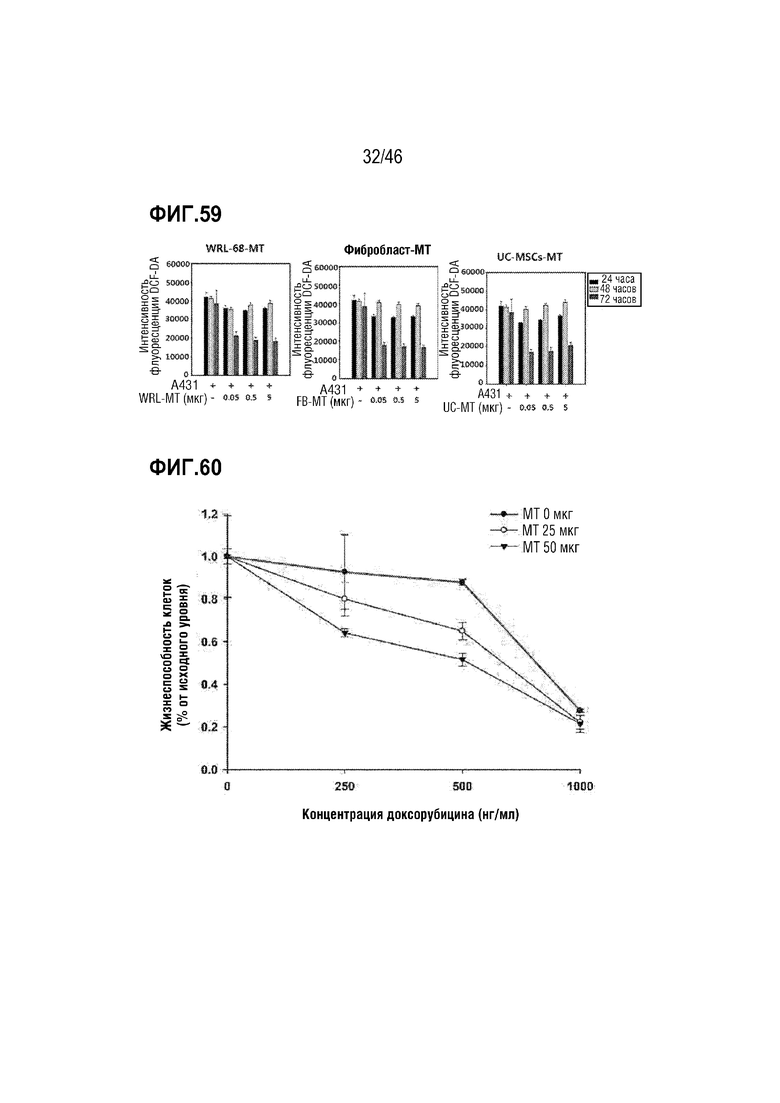
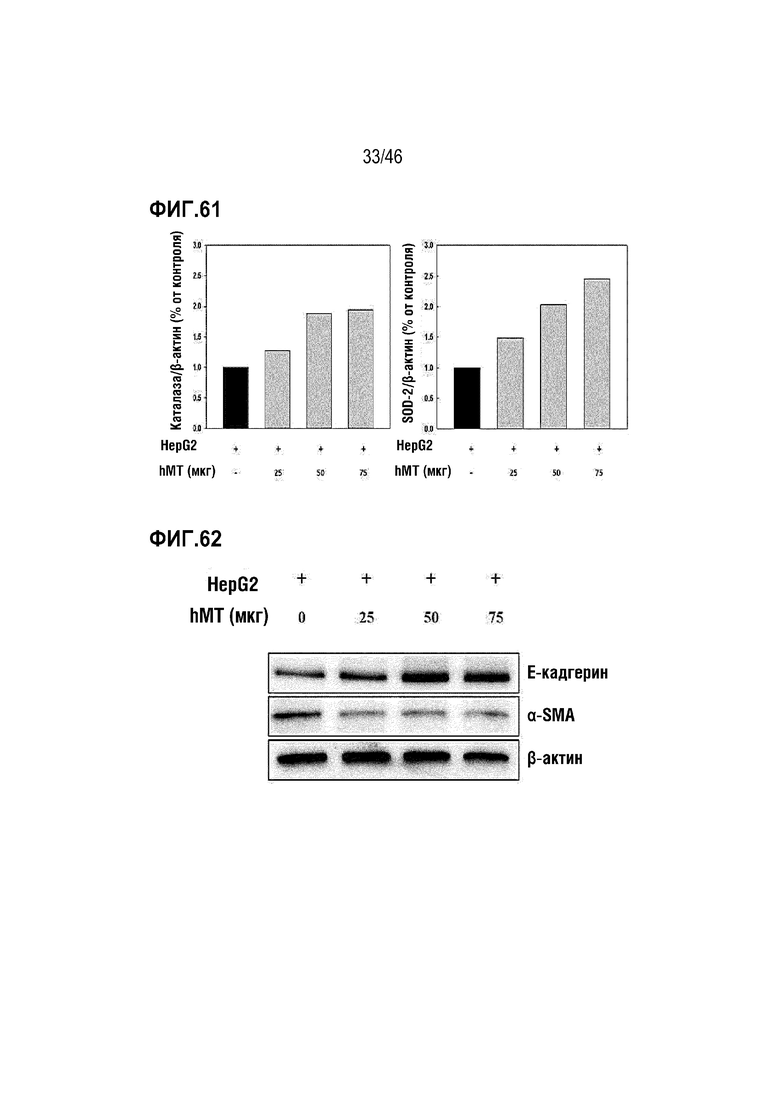

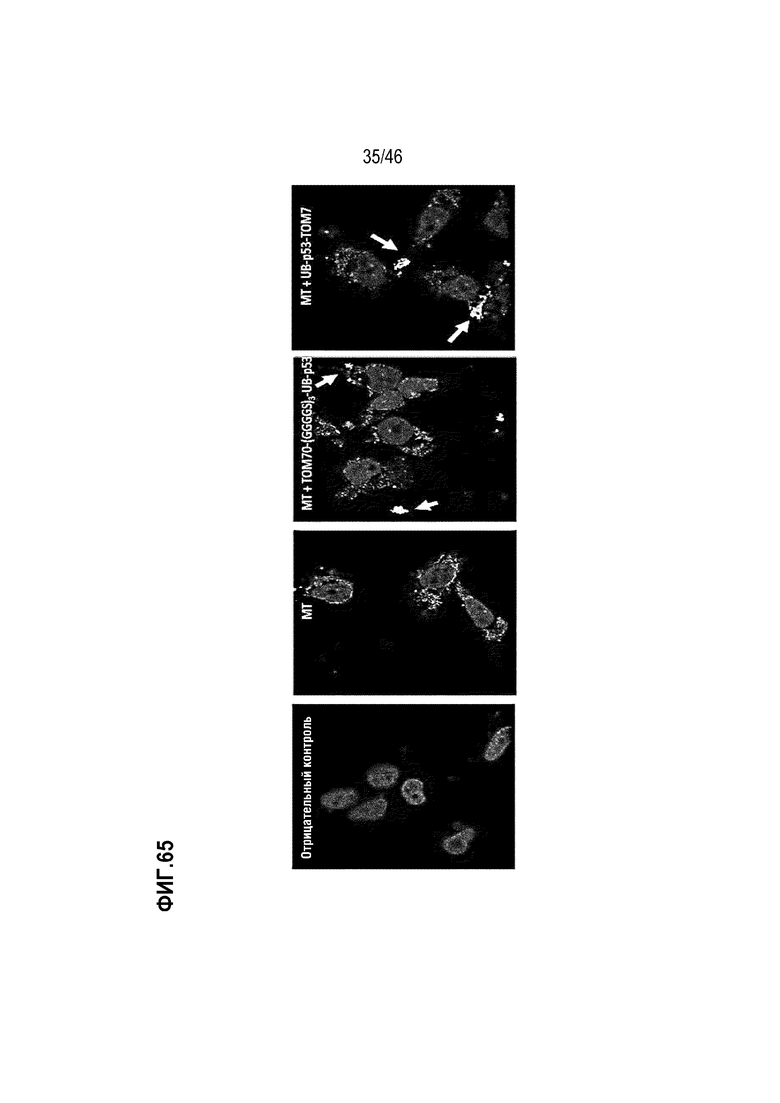


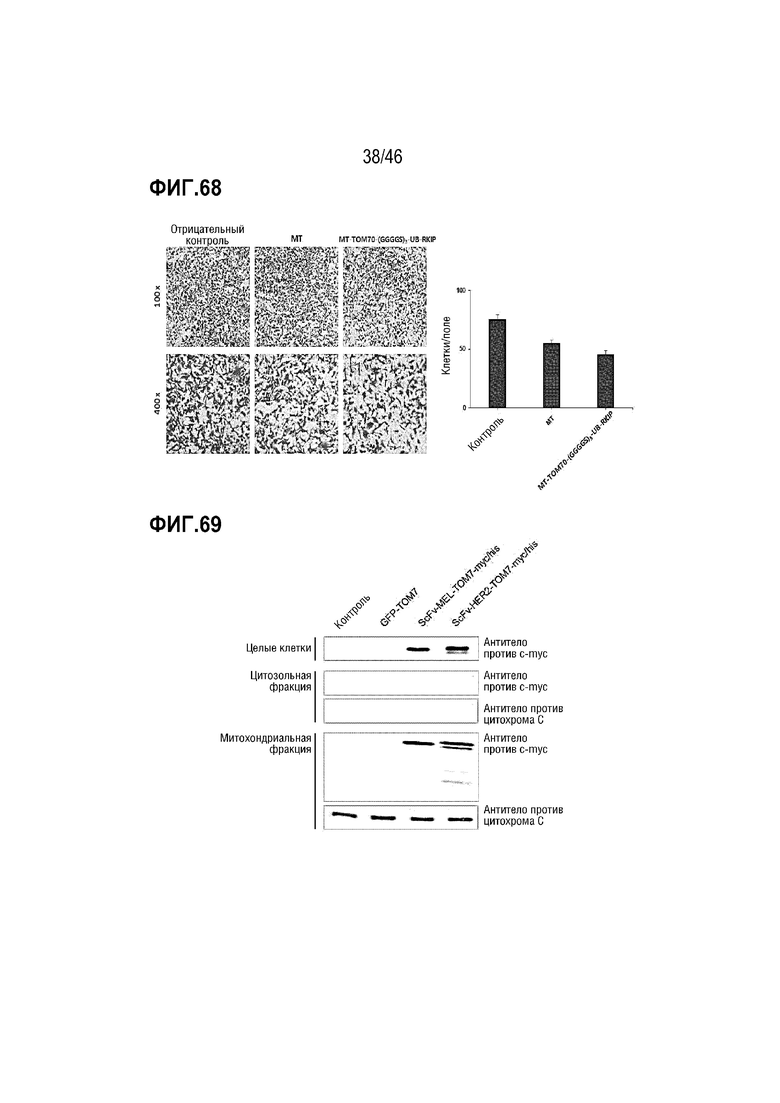
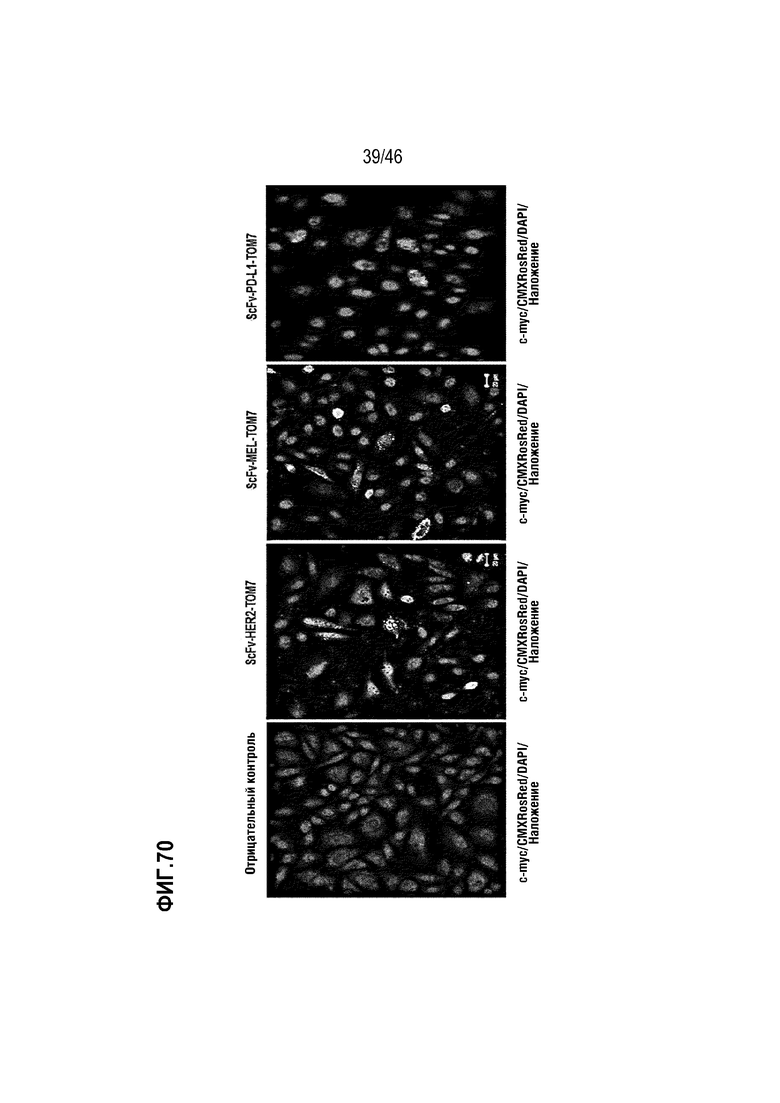
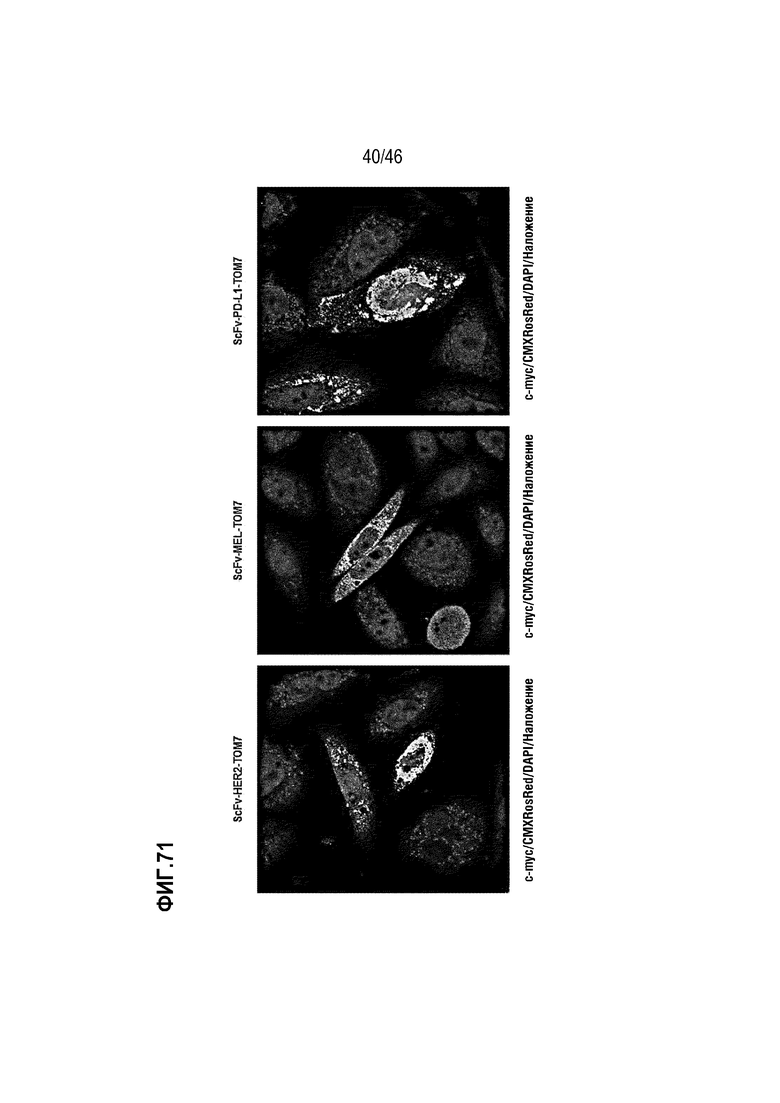
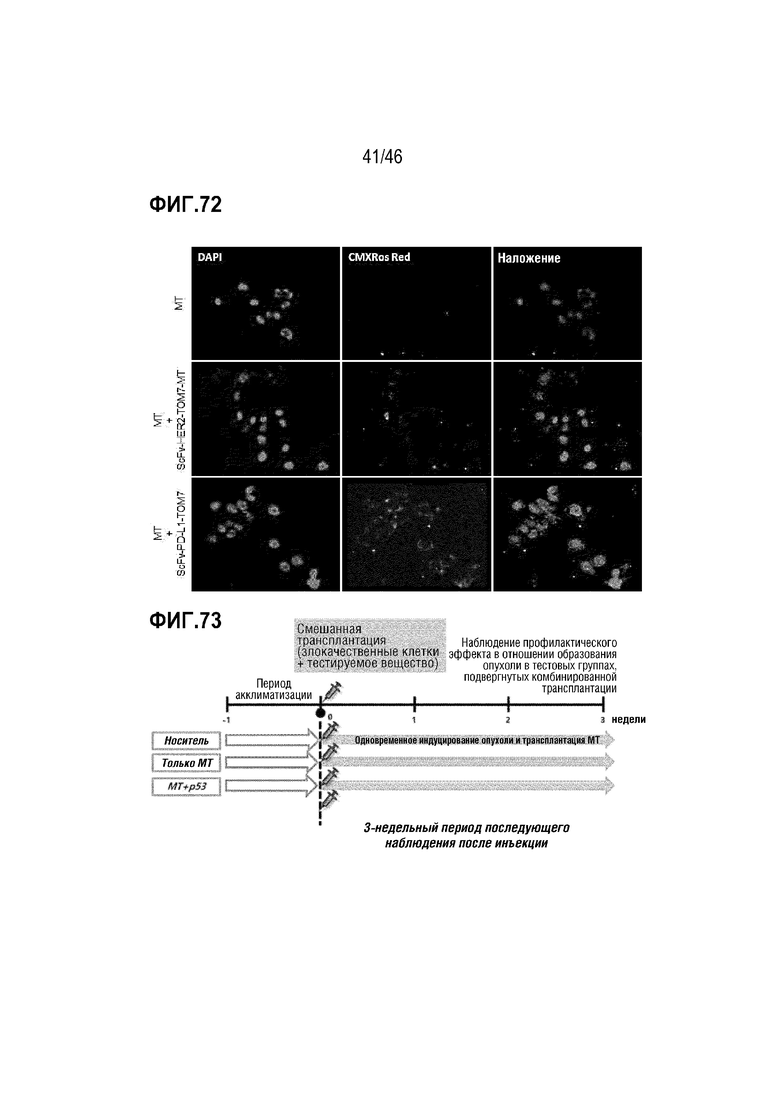
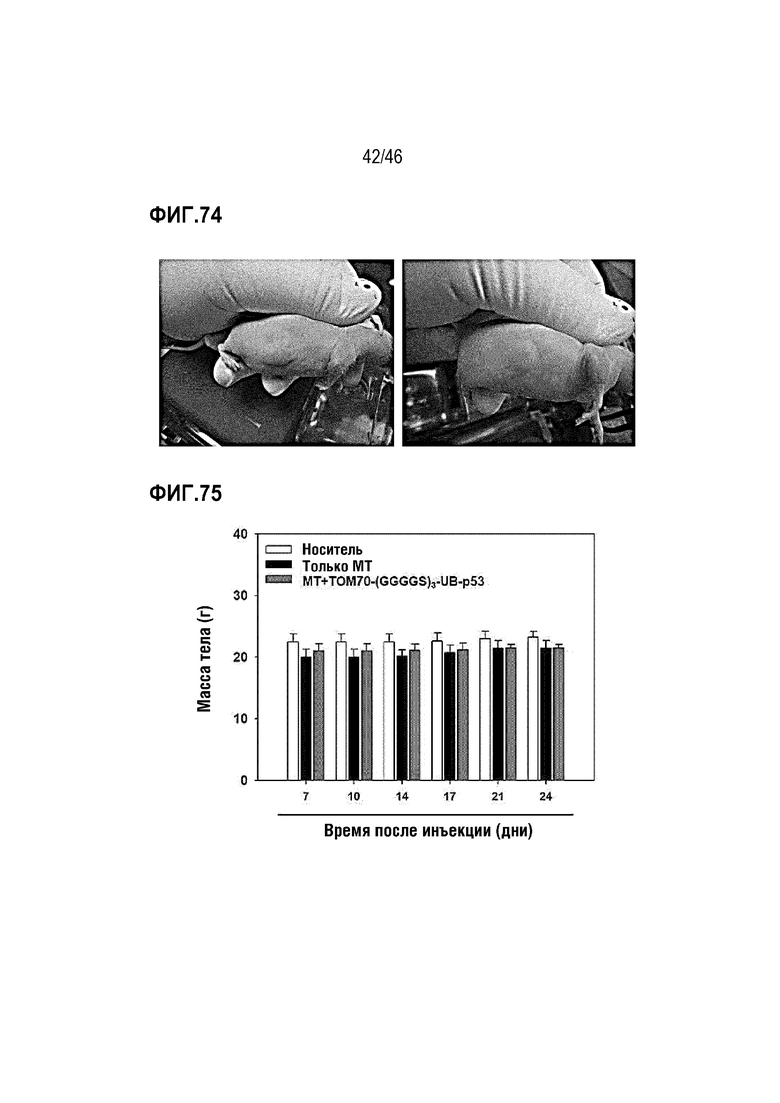


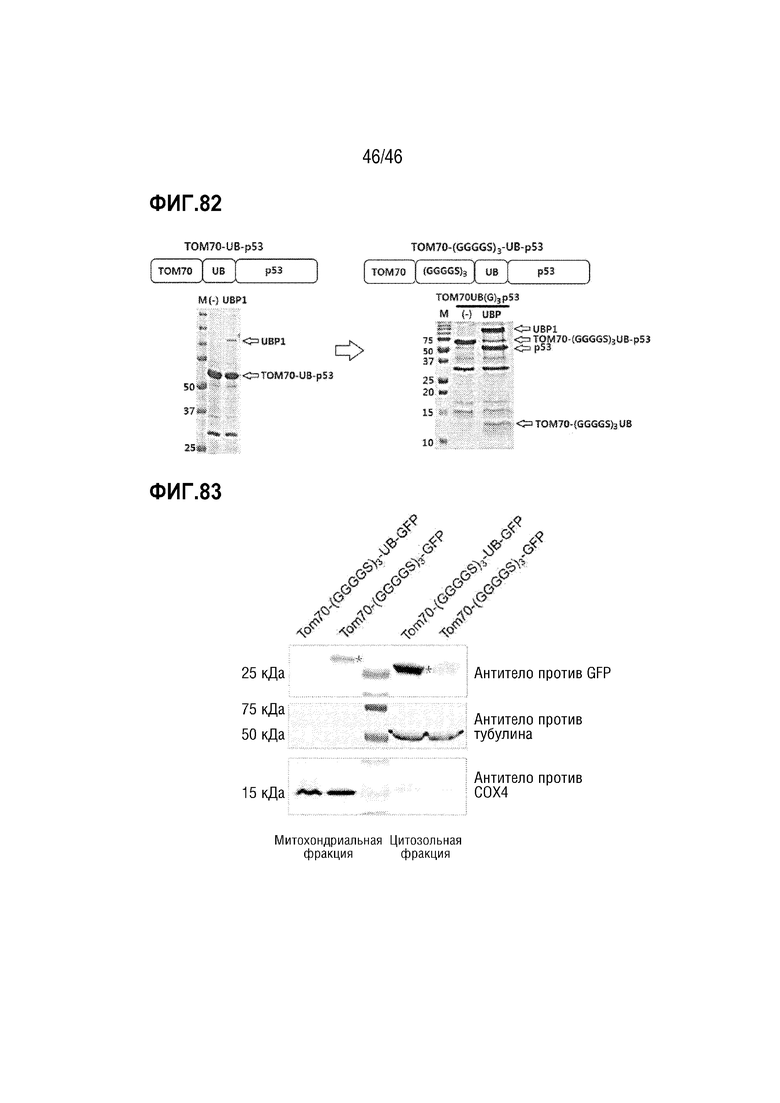
Изобретение относится к молекулярной биологии, биотехнологии и медицине, в частности к митохондриям, модифицированным посредством слитого белка, и фармацевтической композиции, содержащей их в качестве активного ингредиента. Митохондрии по настоящему изобретению можно эффективно доставлять к мишени, в клетку, где они проявляют различные эффекты, или в организм. Модифицированные митохондрии можно использовать в качестве противоопухолевого средства, в лечении различных заболеваний. Кроме того, слитый белок, содержащий интересующий белок, и слитый белок, содержащий белок для таргетинга, можно использовать для модификации митохондрий. Изобретение предоставляет эффективную систему доставки белка с использованием митохондрий в качестве носителя для лечения заболеваний. 6 н. и 24 з.п. ф-лы, 83 ил., 12 табл., 26 пр.
1. Модифицированная митохондрия для доставки чужеродного белка в клетки, в которой чужеродный белок связан с внешней мембраной митохондрий, где чужеродный белок представляет собой слитый белок, содержащий пептид, заякоривающий в митохондриях, и желаемый белок, способный функционировать внутри и вне клетки, или слитый белок, содержащий белок для таргетинга мишени, имеющий способность связываться с лигандом или рецептором, присутствующим в мембране клетки, и пептид, заякоривающий в митохондриальной внешней мембране,
где пептид, заякоривающий в митохондриях, содержит N-концевую область или C-концевую область белка, присутствующего в белке митохондриальной мембраны, и белок, присутствующий в белке митохондриальной мембраны, является любым белком, выбранным из группы, состоящей из TOM20, TOM70, OM45, TOM5, TOM6, TOM7, TOM22, Fis1, Bcl-2, Bcl-x и VAMP1B.
2. Модифицированная митохондрия по п.1, где митохондрия выделена из клеток или тканей.
3. Модифицированная митохондрия по п.2, где клетка является любой клеткой, выбранной из группы, состоящей из соматических клеток, половых клеток, стволовых клеток и их комбинации.
4. Модифицированная митохондрия по п.1, где чужеродный белок связан с внешней мембраной митохондрии с помощью пептида, заякоривающего в митохондриях.
5. Модифицированная митохондрия по п.1, отличающаяся тем, что N-концевая область или C-концевая область белка, присутствующего в белке митохондриальной мембраны, находится на внешней мембране митохондрий.
6. Модифицированная митохондрия по п.1, отличающаяся тем, что заякоривающий пептид содержит N-концевую область любого белка, выбранного из группы, состоящей из TOM20, TOM70 и OM45.
7. Модифицированная митохондрия по п.1, отличающаяся тем, что пептид, заякоривающий в митохондриях, содержит C-концевую область любого белка, выбранного из группы, состоящей из TOM5, TOM6, TOM7, TOM22, Fis1, Bcl-2, Bcl-x и VAMP1B.
8. Модифицированная митохондрия по п.1, где желаемый белок является любым белком, выбранным из группы, состоящей из активного белка, проявляющего активность в клетке, белка, присутствующего в клетке, и белка, имеющего способность связываться с лигандом или рецептором, присутствующим в мембране клетки.
9. Модифицированная митохондрия по п.8, где желаемый белок является любым белком, выбранным из группы, состоящей из p53, гранзима B, Bax, Bak, PDCD5, E2F, AP-1 (Jun/Fos), EGR-1, белка ретинобластомы (RB), гомолога фосфатазы и тензина (PTEN), E-кадгерина, нейрофибромина-2(NF-2), поли-АДФ-рибоза-синтазы 1 (PARP-1), BRCA-1, BRCA-2, белка аденоматозного полипоза толстой кишки (APC), фактора, ассоциированного с рецептором фактора некроза опухоли (TRAF), ингибиторного белка RAF-киназы (RKIP), p16, KLF-10, LKB1, LHX6, C-RASSF, DKK-3PD1, Oct3/4, Sox2, Klf4 и c-Myc.
10. Модифицированная митохондрия по п.1, где чужеродный белок является желаемым белком, связанным с N-концевой областью TOM20, TOM70 или OM45.
11. Модифицированная митохондрия по п.10, где чужеродный белок связан в следующем порядке:
N-конец-N-концевая область TOM20, TOM70 или OM45-желаемый белок-C-конец.
12. Модифицированная митохондрия по п.11, где чужеродный белок дополнительно содержит аминокислотную последовательность, распознаваемую протеолитическим ферментом в эукариотических клетках, или убиквитин или его фрагмент между заякоривающим пептидом и желаемым белком.
13. Модифицированная митохондрия по п.12, где фрагмент убиквитина содержит C-концевые Gly-Gly аминокислотной последовательности SEQ ID NO: 71 и содержит от 3 до 75 последовательных аминокислот с C-конца.
14. Модифицированная митохондрия по п.12, где чужеродный белок дополнительно содержит линкер между желаемым белком и убиквитином или его фрагментом.
15. Модифицированная митохондрия по п.14, где линкер представляет собой полипептид SEQ ID NO: 70.
16. Модифицированная митохондрия по п.8, где белок, имеющий способность связываться с лигандом или рецептором, присутствующим в мембране клетки, является лигандом или рецептором, присутствующим на поверхности опухолевой клетки.
17. Модифицированная митохондрия по п.16, где лиганд или рецептор, присутствующий на поверхности опухолевой клетки, является любым лигандом или рецептором, выбранным из группы, состоящей из CD19, CD20, меланома-ассоциированного антигена E (MAGE), NY-ESO-1, карциноэмбрионального антигена (CEA), муцина 1, ассоциированного с поверхностью клетки (MUC-1), простатической кислой фосфатазы (PAP), простат-специфического антигена (PSA), сурвивина, тирозиназа-связанного белка 1 (tyrp1), тирозиназа-связанного белка 1 (tyrp2), гена брахиурии, мезотелина, рецептора эпидермального фактора роста(EGFR), рецептора эпидермального фактора роста 2 человека (HER-2), ERBB2, белка опухоли Вильмса (WT1), FAP, EpCAM, PD-L1, ACPP, CPT1A, IFNG, CD274, FOLR1, EPCAM, ICAM2, NCAM1, LRRC4, UNC5H2 LILRB2, CEACAM, нектина-3 или их комбинации.
18. Модифицированная митохондрия по п.1, где чужеродный белок связан с C-концевой областью любого белка, выбранного из группы, состоящей из TOM5, TOM6, TOM7, TOM22, Fis1, Bcl-2, Bcl-x и VAMP1B.
19. Модифицированная митохондрия по п.18, где чужеродный белок связан в следующем порядке:
N-конец-желаемый белок-C-концевая область любого белка, выбранного из группы, состоящей из TOM5, TOM6, TOM7, TOM22, Fis1, Bcl-2, Bcl-x и VAMP1B-C-конец.
20. Модифицированная митохондрия по п.19, где чужеродный белок дополнительно содержит линкер между желаемым белком и C-концевой областью любого белка, выбранного из группы, состоящей из TOM5, TOM6, TOM7, TOM22, Fis1, Bcl-2, Bcl-x и VAMP1B.
21. Модифицированная митохондрия по п.20, где линкер представляет собой полипептид SEQ ID NO: 70.
22. Фармацевтическая композиция для профилактики или лечения злокачественного новообразования, содержащая модифицированную митохондрию по любому из пп.1-21 в качестве активного ингредиента и буферный раствор,
где злокачественное новообразование является любым злокачественным новообразованием, выбранным из группы, состоящей из рака желудка, рака печени, рака легких, колоректального рака, рака молочной железы, рака предстательной железы, рака яичников, рака поджелудочной железы, рака шейки матки, рака щитовидной железы, рака гортани, острого миелолейкоза, опухоли головного мозга, нейробластомы, ретинобластомы, рака головы и шеи, рака слюнных желез и лимфомы.
23. Применение модифицированной митохондрии по пп 1-21 для профилактики или лечения злокачественного новообразования,
где злокачественное новообразование является любым злокачественным новообразованием, выбранным из группы, состоящей из рака желудка, рака печени, рака легких, колоректального рака, рака молочной железы, рака предстательной железы, рака яичников, рака поджелудочной железы, рака шейки матки, рака щитовидной железы, рака гортани, острого миелолейкоза, опухоли головного мозга, нейробластомы, ретинобластомы, рака головы и шеи, рака слюнных желез и лимфомы.
24. Применение модифицированной митохондрии по любому из пп. 1-21 в качестве средств внутриклеточной и внеклеточной доставки чужеродного белка, содержащего желаемый белок, способный функционировать вне и внутри клетки.
25. Применение модифицированной митохондрии по п.24, где чужеродный белок содержит пептид, заякоривающий в митохондриальной внешней мембране, и связан с внешней мембраной митохондрий посредством пептида, заякоривающего во внешней мембране, и его доставляют внутрь и вне клетки.
26. Модифицированная митохондрия по п.1, где белок для таргетинга мишени, имеющий способность связываться с лигандом или рецептором, присутствующим в мембране клетки, и пептид, заякоривающий в митохондриальной внешней мембране, связаны от N-конца к C-концу.
27. Модифицированная митохондрия по п.26, где белок для таргетинга мишени, имеющий способность связываться с лигандом или рецептором, присутствующим в мембране клетки, является антителом или его фрагментом.
28. Модифицированная митохондрия по п.27, где фрагмент антитела является любым фрагментом, выбранным из группы, состоящей из Fab, Fab', scFv и F(ab)2.
29. Способ получения модифицированной митохондрии по п. 1, включающий стадию смешивания выделенных митохондрий с чужеродным белком, где чужеродный белок представляет собой слитый белок, содержащий пептид, заякоривающий в митохондриях, и желаемый белок, способный функционировать внутри и вне клетки, или слитый белок, содержащий белок для таргетинга мишени, имеющий способность связываться с лигандом или рецептором, присутствующим в мембране клетки, и пептид, заякоривающий в митохондриальной внешней мембране,
где пептид, заякоривающий в митохондриях, содержит N-концевую область или C-концевую область белка, присутствующего в белке митохондриальной мембраны, и белок, присутствующий в белке митохондриальной мембраны, является любым белком, выбранным из группы, состоящей из TOM20, TOM70, OM45, TOM5, TOM6, TOM7, TOM22, Fis1, Bcl-2, Bcl-x и VAMP1B.
30. Способ получения модифицированной митохондрии по п. 1 из трансформированных клеток посредством инъецирования полинуклеотида, кодирующего чужеродный белок, где чужеродный белок представляет собой слитый белок, содержащий пептид, заякоривающий в митохондриях, и желаемый белок, способный функционировать внутри и вне клетки, или слитый белок, содержащий белок для таргетинга мишени, имеющий способность связываться с лигандом или рецептором, присутствующим в мембране клетки, и пептид, заякоривающий в митохондриальной внешней мембране,
где пептид, заякоривающий в митохондриях, содержит N-концевую область или C-концевую область белка, присутствующего в белке митохондриальной мембраны, и белок, присутствующий в белке митохондриальной мембраны, является любым белком, выбранным из группы, состоящей из TOM20, TOM70, OM45, TOM5, TOM6, TOM7, TOM22, Fis1, Bcl-2, Bcl-x и VAMP1B.
| WO 2011058408 A2, 19.05.2011 | |||
| WO 2015120296 A1, 13.08.2015 | |||
| MOSSALAM M | |||
| et al., Direct induction of apoptosis using an optimal mitochondrially targeted p53, Mol Pharm, 2012, vol | |||
| Разборный с внутренней печью кипятильник | 1922 |
|
SU9A1 |
| Кипятильник для воды | 1921 |
|
SU5A1 |
| Способ извлечения сульфо-нефтяных или т.п. кислот | 1923 |
|
SU1449A1 |
| YANG Y.W., KOOB M.D., Transferring isolated mitochondria into tissue culture cells, Nucleic Acids Res, 2012, vol | |||
| Приспособление с иглой для прочистки кухонь типа "Примус" | 1923 |
|
SU40A1 |
| Способ изготовления электрических сопротивлений посредством осаждения слоя проводника на поверхности изолятора | 1921 |
|
SU19A1 |
| ВЕРЕЩАГИНА | |||
Авторы
Даты
2024-01-12—Публикация
2019-04-25—Подача