Настоящее изобретение относится к области моделирования молекулярных взаимодействий для оценки заболеваний и терапии заболеваний. В частности, изобретение относится к системе моделирования молекулярных взаимодействий, принимающих участие в воспалительном процессе у субъекта, где указанная система включает блок обработки данных, включающий множество наборов данных, каждый из которых включает по меньшей мере один идентификатор для молекулы, которая может принимать участие в патологическом процессе, данные по молекулярным взаимодействиям указанной молекулы с одной или более другими молекулами и по меньшей мере одну характеристику данных, которая предназначена для набора данных, причем каждый набор данных по меньшей мере в одном отношении связан с другим набором данных на основании молекулярных взаимодействий, при этом наборы данных сгруппированы в секции данных с идентичными характеристиками данных, и где характеристики данных указывают на биологическую функцию молекулы в воспалительном процессе и алгоритм компьютерной программы включен в блок обработки данных, который генерирует карту сети, основанную на множестве наборов данных в базе данных и который позволяет идентифицировать узлы в карте сети на основании предварительно определенных параметров, и блок визуализации, который позволяет определять молекулярные взаимодействия в идентифицированных узлах. Кроме того, в настоящем изобретении предлагается способ моделирования молекулярных взаимодействий, принимающих участие в воспалительном процессе у субъекта, а также применение системы по настоящему изобретению для моделирования молекулярных взаимодействий, принимающих участие в воспалительном процессе.
Воспаление означает ответную реакцию организма на неблагоприятные факторы, такие как патогены, повреждение или физические и химические раздражающие факторы, приводящие к повреждению клеток. Защитные средства включают клетки иммунной системы и кровеносные сосуды, которые служат для удаления некротических клеток и поврежденных тканей, а также для стимуляции восстановления ткани.
Воспаление является сложным биологическим процессом, который можно разделить на острые воспалительные процессы и хронические воспалительные процессы. Основные симптомы воспаления включают боль, повышенную температуру тела, покраснение, отечность и функциональные нарушения.
Острое воспаление обычно включает резидентные иммунные клетки, присутствующие в пораженной ткани. На этих клетках присутствуют поверхностные рецепторы, известные как образ-распознающие рецепторы (PRR), которые распознают два подкласса молекул: патоген-ассоциированные образ-распознающие молекулы (РАМР) и ассоциированные с повреждением образ-распознающие молекулы (DAMP). РАМР являются патоген-специфическими молекулами, a DAMP являются молекулами, ассоциированными с повреждением организма и клеток хозяина.
В начале развития воспаления молекулы PRR распознают РАМР или DAMP и индуцируют воспалительные медиаторы, ответственные за клинические симптомы воспаления, а также несколько клеточных и внеклеточных биохимических каскадов, распространяющих воспалительную ответную реакцию (см. например, Netea 2017).
Следует понимать, что лекарственные средства, принимающие участие в лечении различных аспектов воспаления, могут действовать на различных уровнях регуляции комплексных молекулярных путей, включенных в различные фазы воспаления. Таким образом, является перспективным исследование моделирования молекулярных процессов, принимающих участие в воспалительном процессе, для того, чтобы можно было предсказать действие лекарственного средства и результат терапии.
Недавно сообщалось о моделях in silico, которые способны моделировать комплексные молекулярные взаимодействия в ходе геномного и транскриптомного анализа (Wolkenhauer 2002, Steffen 2017). Более того, для различных типов рака были созданы сети регуляторных генов и моделирования заболеваний (см. Sadeghi 2016, Dryer 2018 и Khan 2018).
Однако пока отсутствует информация о моделировании воспалительного процесса, которая могла бы ускорить анализ in silico воспалительных процессов и разработку лекарственных средств, и в настоящее время все еще существует настоятельная потребность в такой информации.
Таким образом, техническая проблема, лежащая в основе настоящего изобретения, заключается в обеспечении средств и способов для удовлетворения вышеупомянутых потребностей. Эта задача решается с помощью вариантов осуществления, охарактеризованных здесь в формуле изобретения и далее в описании.
Следовательно, в настоящем изобретении предлагается система для моделирования молекулярных взаимодействий, принимающих участие в воспалительном процессе у субъекта, включающая:
(I) блок обработки данных, включающий:
(а) базу данных, включающую множество наборов данных, каждый из которых включает:
(i) по меньшей мере один идентификатор молекулы, для которой предполагается участие в патологическом процессе,
(ii) данные по молекулярным взаимодействиям указанной молекулы с одной или более других молекул, и
(iii) по меньшей мере одну характеристику данных, выделенную для набора данных, где каждый набор данных связан по меньшей мере в одном отношении с другим набором данных в базе данных, основанной на молекулярных взаимодействиях,
где наборы данных сгруппированы в секции данных, включающие наборы данных, содержащих идентичные характеристики данных; и
где характеристики данных указывают на биологическую функцию молекулы в воспалительном процессе;
и
(б) алгоритм на основе компьютерной программы, включенный в блок обработки данных, который генерирует карту сети, основанную на множестве наборов данных в базе данных и который позволяет идентифицировать узлы в карте сети на основании предварительно определенных параметров; и
(II) блок визуализации, который позволяет определять молекулярные взаимодействия, принимающие участие в воспалительном процессе, в идентифицированных узлах.
Термин «система», использованный здесь, относится к любому модулю, содержащему (I) блок обработки и (II) блок визуализации, как описано выше. Следует понимать, что каждый из упомянутых блоков может также состоять из нескольких отдельных устройств или подблоков. Например, блок обработки может содержать запоминающее устройство для базы данных, содержащее множество наборов данных, упомянутых выше, и процессор, реализующий алгоритм на основе компьютерной программы, который генерирует карту сети на основе множества наборов данных в базе данных и который позволяет идентифицировать узлы на карте сети на основе предварительно определенных параметров. Более того, система может включать устройство для ввода данных, такое как устройство для автоматического ввода данных, например, считывающее устройство или приемное устройство для данных из других устройств обработки или интернета, или устройство ля ввода данных вручную. Предпочтительно система может также включать компоненты для передачи данных между индивидуальными блоками, подблоками и/или устройствами. Такую передачу данных можно осуществлять с помощью постоянного или временного физического соединения, такого как коаксиальный, оптоволоконный, волоконно-оптический кабель или кабель парной скрутки, кабели 10 BASE-T. В другом варианте передачу данных можно осуществлять с помощью временного или постоянного беспроводного соединения с использованием, например, радиоволн, таких как Wi-Fi, LTE, LTE с расширенными возможностями или Bluetooth. Блоки и другие компоненты системы могут быть расположены в непосредственной близости или могут быть физически отделены друг от друга.
Термин «моделирование», использованный здесь, означает установку виртуальной модели молекулярных взаимодействий, принимающих участие в воспалительном процессе в системе по настоящему изобретению. Такое моделирование, предпочтительно, представляет собой динамическое моделирование, то есть при этом учитываются изменения взаимодействий в течение времени и в ответ на моделируемые внешние воздействия. Более того, моделирование может быть детерминированным моделированием, исключающим статистические события, или может включать такие события.
Термин «молекулярные взаимодействия, принимающие участие в воспалительном процессе», относится к описанным здесь физиологическим и патофизиологическим взаимодействиям между биологическими молекулами, которые, как известно, присутствуют в организме субъекта и принимают участие в воспалении. Термин «воспаление», использованный здесь, обычно означает патофизиологическую ответную реакцию субъекта на неблагоприятные факторы, такие как, например, патогены, повреждение клеток или раздражающие факторы. Воспаление может представлять собой острое или хроническое воспаление. Предпочтительно, воспаление согласно настоящему изобретению означает хроническое воспаление. Типичными признаками воспаления являются жар, боль, покраснение или другие кожные реакции, отек и нарушение функции тканей. Воспаление, как правило, начинается когда присутствующие в ткани клетки врожденной иммунной системы обнаруживают инфекцию или повреждение в ткани. Секреция химических сигнальных молекул, таких как хемокины и цитокины, приводит к мобилизации циркулирующих нейтрофилов к участку повреждения или инфекции. Переключение класса липидных медиаторов происходит, когда нейтрофилы накапливаются в гное или гнойном экссудате. Липоксины (LX) стимулируют приток нефлогистических моноцитов. LX, резолвины (Rv) и другие специализированные про-разрешающие медиаторы (SPM), продуцируются в гное, чтобы ограничить или остановить дальнейшую инфильтрацию нейтрофилов в ткани. Каждый из SPM, Rv, марезины (MaR) индуцирует эффероцитоз апоптотических нейтрофилов и клеточный дебрис макрофагами. Разрешающие макрофаги и апоптотические нейтрофилы также продуцируют SPM. При отеке также происходит перенос циркулирующих n-3 полиненасыщенных жирных кислот (PUFA) в экссудаты для временного преобразования в SPM клетками экссудата. Марезины (MaR) и специфические Rv усиливают ранозаживление и регенерацию тканей. Впоследствии гомеостаз тканей может восстанавливаться. Существуют различные воспалительные заболевания, при которых устранение воспаления явно нарушено.
Молекулы, принимающие участие в молекулярных взаимодействиях, которые необходимо моделировать согласно настоящему изобретению, можно идентифицировать с использованием ряда методов. Обычно предварительно определенные молекулы, о которых известно, что они принимают участие в воспалении, или молекулы, о которых известно, что они ассоциированы с предварительно определенными признаками воспалительного процесса, используются в качестве первого набора молекул (также в дальнейшем называемых «затравочные молекулы»), который должен быть включен в базу данных системы по настоящему изобретению.
В случае воспаления затравочными молекулами на первом этапе предпочтительно являются молекулы из ассоциированных с повреждением образ-распознающих молекул (DAMP) и/или из патоген-ассоциированных образ-распознающих молекул (РАМР). Молекулы могут представлять собой белки, пептиды, нуклеиновые кислоты, такие как РНК и ДНК, и/или биологические молекулы, такие как липиды или метаболиты. Молекулы, принимающие участие в молекулярных взаимодействиях, особенно молекулы DAMP или РАМР, предпочтительно идентифицируют при оценке научных данных. В другом варианте молекулы можно идентифицировать при оценке исследований, которые направлены на идентификацию таких молекул. Как правило, доступные научные данные, например, в научных базах данных, таких как PubMed или OMIM, можно оценивать с помощью алгоритмов автоматического анализа текстовой информации, которые позволяют идентифицировать молекулы, предположительно принимающие участие в воспалении, то есть с помощью поиска и анализа литературных данных. Пригодные алгоритмы для идентификации молекул, принимающих участие в воспалении, могут идентифицировать ключевые слова и/или ключевые параметры в научных данных, которые ассоциированы с ролью в воспалении. Предпочтительным способом идентификации молекул, которые принимают участие в молекулярных взаимодействиях при воспалении, является способ описанный в Khan et al (Khan, 2018). Подробное описание идентификации таких затравочных молекул представлено ниже в прилагаемых примерах.
DAMP-ассоциированные молекулы предпочтительно выбирают на первой стадии из группы, состоящей из высокомобильной группы бокса 1, гиалуронан глюкозаминогликана, гепаран сульфата, мочевой кислоты, интерлейкина (IL)-1α, интерлейкина (IL)-1β, интерлейкина 16, интерлейкина 18, фактора роста фибробластов, галектин-3, галектин-1, эндотелиального активирующего моноциты полипептида-II, фактора ингибирующего миграцию макрофагов, белков семейства NLR, содержащих три домена: пирин, PYD и CARD, цистеин-аспарагиновой кислоты-протеазы 1, сшитого димера рибосомального белка S19, лизофосфатидилхолина, внутриклеточной, ассоциированной с мембраной, кальций-независимой фосфолипазой А2 бета, тирозил-TRNA синтетазы, белка S100-A8, белка S100 А-9 и кальретикулина.
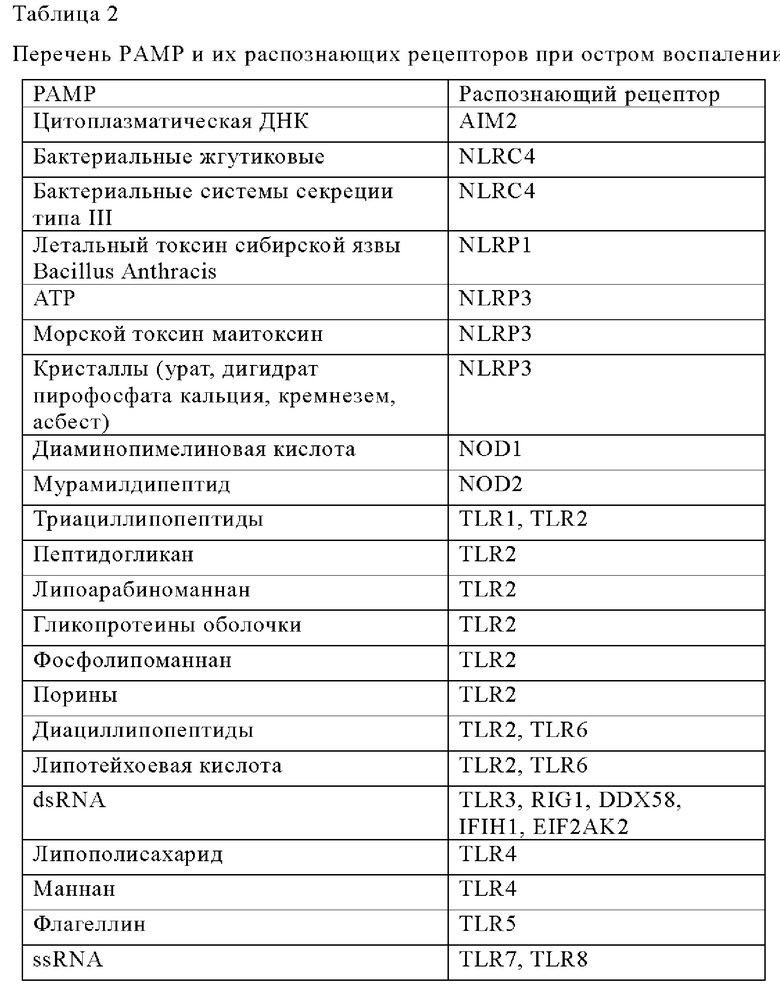
На первой стадии РАМР-ассоциированные молекулы предпочтительно выбирают из группы, состоящей из цитоплазматической ДНК, бактериальных жгутиковидных, бактериальных секреторных систем типа III, бактериального токсина сибирской язвы Bacillus Anthracis, ATP, майтотоксина из морских организмов, кристаллов (ураты, дигидрат пирофосфата кальция, кремнезем, асбест), диаминопимелиновой кислоты, мурамиллипептиды, триацил-липопептидов, пептидогликана, липоарабиноманнана, гликопротеинов оболочки, фосфолипоманнана, поринов, диациллипопептидов, липотейхоевой кислоты, dsRNA (двухцепочечная РНК), липополисахарида, маннана, флагеллина, ssRNA (одноцепочечная РНК) и их распознающих рецепторов. Распознающие рецепторы представляют собой AIM2, NLRC4, NLRP1, NLRP3, NODI, TLR1, TKR2, TLR6, TLR3, RIG1, DDX58, IFIH1, EIF2AK2, TLR4, TLR5, TLR7 и TLR8.
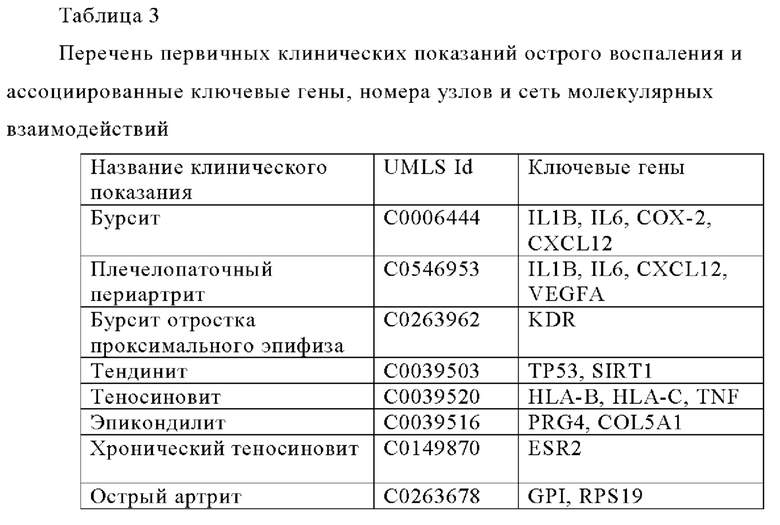
На еще одной стадии можно идентифицировать дополнительные затравочные молекулы с помощью оценки научных данных, относящихся к симптомам острого воспаления с использованием поиска и анализа литературных данных, цель которых заключается в идентификации молекул, ассоциированных с указанными признаками острого воспаления. Обычно, клинические симптомы острого воспаления выбирают из группы, состоящей из гонококкового артрита, вирусного артрита, тендонита, бурсита, острой травмы позвоночного диска, мышечной травмы/разрыва мышц, травмы мягких тканей, эпифизарных травм, травмы в результате перенапряжения, растяжения связок голеностопного сустава, травмы таранно-малоберцовой связки, медиального ушиба бедренного сустава, тендонита и бурсита шейки бедра, ушибов, травмы медиальной коллатеральной связки колена, травмы задней крестообразной связки колена, синдрома подвздошно-большеберцового тракта, травмы акромиально-ключичного сустава, травмы мышц-вращателей плеча, тендинита надостной мышцы, травмы при растяжении/напряжении шейного отдела позвоночника, травмы крестцово-подвздошной связки, тендонита бицепсной мышцы, латерального эпикондилита, медиального эпикондилита, вывиха и растяжения шейного отдела позвоночника, разрыва ахиллова сухожилия, пяточного бурсита, растяжения мышцы задней поверхности бедра или разрыва бицепсной мышцы. Более предпочтительно, для дополнительных затравочных молекул, принимающих участие в воспалении, оценивают по меньшей мере следующие острые клинические показания: бурсит, субакромиальный бурсит, бурсит локтевого отростка, тендинит, тендосиновит, эпикондилит, болезнь де Кервена, и острый артрит. Обычно процесс поиска и анализа литературных данных включает автоматический скрининг научной литературы в базах данных, таких как PubMed и/или OMIM, и скрининг баз данных, ассоциированных с заболеваниями-генами, таких как DisGeNET, DISEASE, KEGG, предназначенных для поиска генов, ассоциированных с клиническим показанием с использованием алгоритмов текстового поиска и анализа. Более подробное описание идентификации таких затравочных молекул представлено ниже в прилагаемых примерах.
Дополнительные затравочные молекулы предпочтительно включают IL1b, IL6, СОХ-2, CXCL12, VEGF-A, KDR, ТР53, SIRT1, HLA-b, HLA-C, TNF, PRG4, COL5A1, ESR2, GPI и RS19.
Для указанных выше затравочных молекул была создана база данных в системе по настоящему изобретению. Указанная база данных должна включать множество наборов данных для каждой затравочной молекулы, где каждый набор данных включает:
(i) по меньшей мере один идентификатор для молекулы, которая как предполагается принимает участие в патологическом процессе. Обычно можно использовать научное обозначение или название молекулы. В другом варианте можно использовать уникальный числовой идентификатор или уникальный произвольный идентификатор, который на следующем этапе может быть однозначно отнесен к научному обозначению или названию молекулы. Более предпочтительно, чтобы для каждой затравочной молекулы, как правило, были включены ее конкретные идентификационные номера UNIPROT-, HGNC-, RefSeq-, Ensemble-, PubMed- и NCBI-ID, CHEBI-ID, а также общие альтернативные названия и полное название кодируемого белка.
(ii) данные по молекулярным взаимодействиям указанной молекулы с одной или более другими молекулами. Как правило, следует включать компонент молекулярного взаимодействия или данные, относящиеся к молекулярным регуляторам затравочных молекул. Более предпочтительно такие данные могут включать информацию об источнике и мишени молекул, о регуляторах, таких как ферменты, молекулах, контролирующих экспрессию, таких как транскрипционные факторы или регуляторные РНК и т.п. Кроме того, данные могут также включать информацию о типе модификации (например, катализ, фосфорилирование), о состоянии молекул (например, фосфорилированная форма), о типе молекул (белки, микроРНК, комплексы, ДНК, простые молекулы) и/или о типе взаимодействия (положительный, отрицательный). Такую информацию можно получить автоматически по алгоритму поиска и анализа, такому как приложение BisoGenet арр, доступное на сайте Cytoscape4.0.
(iii) по меньшей мере одну характеристику данных, которая выделена для набора данных. Характеристика данных должна указывать на биологическую функцию затравочной молекулы так, что молекула на основании указанной информации может быть отнесена к функциональной секции воспалительного процесса. Функциональную секцию воспалительного процесса предпочтительно выбирают из группы, состоящей из регуляции гемопоэза, стимуляции врожденного иммунного ответа, передачи сигнала распознающего образ рецептора, регуляции приобретенного иммунного ответа, опосредованного Т-клетками иммунного ответа, регуляции секреции иммуноглобулинов, дегрануляции тучных клеток, селекции Т-клеток, дифференциации иммунных клеток, дифференциации лимфоцитов, миелоидной дифференциации, регуляции пролиферации лимфоцитов, регуляции пролиферации В-клеток и регуляции пролиферации Т-клеток.
Указанную выше информацию, предназначенную для включения в наборы данных для затравочных молекул, можно также идентифицировать при оценке научных данных, которые обычно доступны в базах данных по взаимодействию компонентов или регуляторов. В основном, можно рассматривать экспериментально обоснованные молекулярные мишени из баз данных DIP, BioGrid, HPRD, IntAct, MINT, BIND и String. Более того можно использовать несколько экспериментально подтвержденных регуляторных уровней, которые могут включать микроРНК из баз данных mirBase, miRTarBase, TriplexRNA; факторов транскрипции из баз данных TRNSFAC, TRRUST и HTRIdb; lnc RNAs из баз данных IncRInter, EVLncRNAs, IncRNADisease. Уже доказано, что метаболизм жирных кислот играет важную роль в биосинтезе специфических про-разрешающих молекул (включая резолвины, протектины и марезины). Следовательно, можно также собирать и включать информацию по биосинтезу этих медиаторов разрешения воспаления, наряду со всеми ассоциированными ферментами из базы данных Reactome и опубликованной научной литературы. Идентификация таких затравочных молекул более подробно описана ниже в прилагаемых примерах.
Вышеупомянутые наборы данных о затравочных молекулах структурированы в базе данных системы по настоящему изобретению в том смысле, что каждый набор данных имеет по крайней мере одно отношение с другим набором данных в базе данных на основе молекулярных взаимодействий, причем наборы данных сгруппированы в секции данных, содержащих наборы данных, включающих идентичные характеристики данных на основе характеристик данных наборов данных, указывающих на биологическую функцию затравочной молекулы при воспалении, то есть наборы данных по затравочным молекулам сгруппированы в функциональные секции воспалительного процесса. Пригодные алгоритмы для структурирования данных и построения функциональных секций воспалительного процесса включают подключаемый модуль (плагин) CytoScape ClueGO, который использует базу данных GeneOntology (GO). Результаты можно скорректировать вручную, задав параметры, например, диапазон древовидных уровней термина в GO-Hierarchy, р-значение для ассоциации или количество генов на модуль. Чтобы сосредоточить внимание на процессах, связанных с иммунологией, предпочтительно можно использовать базу данных GO «Immunological_process» при первом запуске модуляризации (разбиение на модули). При втором запуске можно использовать более общую базу данных "biological_process". Более подробно идентификация таких затравочных молекул описана в прилагаемых ниже примерах.
Взаимодействия, моделируемые согласно настоящему изобретению, могут быть связаны с биологическими функциями молекул в воспалительном процессе. Такие биологические функции можно выбрать из группы, состоящей из дегрануляции тучных клеток, дифференциации макрофагов, дифференциации миелоидных клеток, дифференциации лимфоцитов, дифференциации иммунных клеток, регуляции гемопоэза, стимуляции врожденного иммунного ответа, путей передачи сигнала рецептора распознавания образов, продуцирования цитокинов, регуляции приобретенного иммунного ответа, опосредованного Т-клетками иммунного ответа, отбора Т-клеток, регуляции пролиферации В-клеток, регуляции секреции иммуноглобулинов, регуляции пролиферации Т-клеток, регуляции пролиферации лимфоцитов, устранения воспаления, регуляции экспрессии генов, модификации белков, развития кровеносных сосудов, иммунного ответ, гемопоэза, развития нейронов и транспорта белков.
Затравочные молекулы распределяются с помощью структуры базы данных согласно вышеупомянутым биологическим функциям, что приводит к формированию структуры функциональных секций, как описано ранее. Кроме того, следует понимать, что в указанные функциональные секции также распределяется дополнительная информация, содержащаяся в наборах данных по взаимодействию затравочных молекул, компонентов и регуляторов, а также другая информация о воспалении.
Использованный здесь термин «блок обработки данных» относится к устройству обработки данных, в котором запускается алгоритм на основе компьютерной программы по настоящему изобретению и которое функционально связано с базой данных системы по настоящему изобретению. Таким образом, блок обработки в системе согласно изобретению сконфигурирован путем реального встраивания алгоритма на основе компьютерной программы для осуществления способа согласно изобретению в упомянутой системе. Устройство обработки может содержать по меньшей мере одну интегральную схему, сконфигурированную для выполнения логических операций. Обычно процессор может также содержать по меньшей мере одну специализированную интегральную схему (ASIC) и/или по меньшей мере одну программируемую логическую интегральную схему (field-programmable gate array FPGA). Специалистам в данной области техники хорошо известно, как функционально соединить базу данных, которая может храниться на временном или постоянном физически интегрированном устройстве хранения данных или которая может быть доступна через удаленное соединение.
Термин «алгоритм на основе компьютерной программы», используемый здесь, согласно машиночитаемым правилам, которые осуществляют алгоритм, способны генерировать сетевую карту затравочных молекул и сопутствующую информацию на основе множества наборов данных в базе данных. Упомянутая сетевая карта затравочных молекул и их компонентов и регуляторов по взаимодействию позволяет идентифицировать определенные узлы в сетевой карте на основе предварительно определенных параметров. Вышеупомянутые узлы являются точками интеграции в сети. Узел характеризуется тем, что затравочная молекула взаимодействует более чем с двумя молекулами. Предпочтительно более двух молекул представляют собой более двух других затравочных молекул. Однако более двух молекул также могут являться молекулами, включенными в базу данных в качестве сопроводительной информации, то есть компонентами или регуляторами по взаимодействию, определенными для данной затравочной молекулы. Применяемый алгоритм обычно включает правила для приоритизации генов, определения степени узла, определения центральности по посредничеству, идентификации мотива, в частности идентификации петель обратной связи или прямой связи и/или определения ассоциации с воспалением.
Использованный здесь термин «карта сети» относится к визуализированной сети затравочных молекул, как представлено в наборах данных в базе данных системы по настоящему изобретению, включая всю или часть сопроводительной информации по взаимодействию компонентов и регуляторов. Обычно, карту сети упоминаемую здесь, можно создать с помощью компьютерных алгоритмов, таких как реализованные в программном обеспечении CellDesigner и/или Cytoscape. Более подробно карта сети описана в известном уровне техники (Khan, 2018). Согласно настоящему изобретению, были разработаны дополнительные пошаговые процедуры, чтобы упорядочить сетевые компоненты и взаимодействия для лучшей визуализации. На первом этапе затравочные молекулы и другие компоненты в модуле структурированы на основе их взаимодействия с другими модулями (то есть межмодульных связей) таким образом, чтобы большинство взаимодействующих стрелок визуально не пересекались друг с другом при графической визуализации. На следующем этапе компоненты сети упорядочиваются на основе их взаимодействия в модуле, а также их взаимодействия с другими модулями карты. На следующем этапе можно решить другие проблемы с удобочитаемостью для таких компонентов, как белки и комплексы, которые имеют множество меж- и внутримодульных связей. Более подробно такие связи представлены ниже на фигурах и в сопутствующих примерах.
Предпочтительно, можно использовать формализм логического моделирования (Boolean) для моделирования динамического системного поведения сети для различных фенотипов в разные моменты времени, таких как фенотипы заболевания и, согласно настоящему изобретению, воспаление. Логические модели не требуют подробных количественных кинетических параметров, что позволяет их использовать для динамического анализа больших регуляторных сетей. Логические функции можно обучить и/или откалибровать с использованием экспериментальных данных, чтобы сделать их специфичными в отношении контекста/типа клетки/фенотипа заболевания. В таких моделях биологические компоненты, такие как гены, белки, белковые комплексы и другие виды, представлены в узлах, связанных с дискретными значениями состояния (0 или 1), а направленные/отмеченные взаимодействия между узлами представлены логическими функциями, состоящими из логических вентилей, определяющих действие, направленное на определение будущего состояния узлов из текущего состояния их регуляторов. Более подробно такие определения представлены ниже в сопутствующих примерах и описаны в известной области техники (см. например, Khan 2018).
Использованный здесь термин «блок визуализации» относится к любому устройству, с использованием которого можно визуально отображать взаимодействия в узлах карты сети. Таким образом, указанный блок визуализации, как правило, содержит устройство для визуального представления карты сети, например, экран или дисплей, или в этом устройстве можно создавать цифровую или бумажную распечатку карты сети. Блок визуализации, как правило, содержит алгоритм на основе компьютерной программы, который графически размещает узлы с высокой связью близко друг к другу и/или который идентифицирует только узлы для графического отображения, которые связаны внутримодулярно. Типичный экран или устройства отображения могут быть основаны на технологиях EL, LC, LED, OLED, AMOLED, плазменных или квантовых точек. Кроме того, блок визуализации предпочтительно должен содержать процессор данных, который позволяет создавать визуализацию сетевой модели на устройстве для визуального представления карты сети. Процессор данных обычно запускается пригодным алгоритмом, реализованным через компьютерный программный код в процессоре данных. Как правило, указанный алгоритм может использовать коммерчески доступные компьютерные программы, такие как Leaflet.js, JavaScript, jQuery, Inkscape и/или Phyton и другие, упомянутые здесь ниже.
Карту сети предпочтительно можно визуализировать в формате удобном для чтения браузером. Более того, в свете исчерпывающей информации, содержащейся в сетевой карте, предпочтительно предусматривается установить масштабируемый формат для карты. Обычно карту сети можно создать в виде интерактивного изображения. Кроме того, алгоритмы, реализованные в программном обеспечении OpenLayer и Google Maps, обычно можно применять для визуализации на основе браузера. В частности, карту сети можно разделить на три слоя. Базовые уровни представляют собой модули на верхнем уровне, подмодули и виды на среднем уровне и виды и взаимодействия на нижнем уровне (подробности смотри также в прилагаемых примерах и фигурах). Чтобы уменьшить вычислительные затраты и требования к локальному интернету, выполняют мозаичное разбиение изображения. Мозаичный метод позволяет разрезать изображения на матрицу меньших изображений одного и того же размера и сохранять их в определенной структуре папок. При этом можно загружать и отображать только просматриваемые в данный момент части карты сети. Скрипты Python обычно можно использовать для мозаичного и многоуровневого разбиения на слои, чтобы проверить, подходит ли экспорт нашей карты CellDesigner для процесса обработки видеоинформации перед отправкой пользователю.
Для визуализации карты сети можно предпочтительно использовать платформу MINERVA [https://minerva.pages.uni.lu]. Можно установить локальный экземпляр MINERVA и протестировать его с картой сети на предмет различных проблем безопасности и надежности. Преимущественно MINERVA предоставляет возможность использовать OpenLayer в качестве базовой библиотеки визуализации для повышения безопасности данных по сравнению с GoogleMaps. Процесс мозаичного разбиения на слои полностью автоматизирован, также поддерживаются базовые операции мыши для масштабирования и прокрутки MIM. Кроме того, пользователь может выбирать узлы для просмотра аннотаций и в то же время подключаться к различным базам данных по лекарственным средствам и химическим соединениям непосредственно из интерфейса MINERVA для дальнейшего анализа сети. Методика применения этих программных инструментов, как правило, хорошо известна специалистам в данной области техники, и ее можно применять без затруднений. Более подробно такие методики представлены ниже в сопутствующих примерах.
Важным шагом для анализа и прогнозирования биомаркеров и кандидатов в терапевтические препараты является обеспечение направления регулировки (например, активация, ингибирование) к границам, соединяющим различные узлы в сети. Таким образом, можно осуществлять подтверждение и/или включение такой информации в наборы данных, лежащих в основе сетевой карты, в частности если необходимо моделировать паталогические состояния. Для перекрестной проверки и/или распределения нормативных указаний можно проверить вручную публикации, связанные с наборами данных, а нормативные указания можно подтвердить или изменить в зависимости от результатов проверки. В дополнение или в другом варианте, можно использовать ресурсы системной биологии, такие как базы данных BioModel, для назначения нормативных указаний подключенным компонентам с помощью автоматизированных сценариев. Многие базы данных используют методы интеллектуального анализа текста, чтобы выделить даже экспериментально подтвержденные взаимодействия с ложноположительной информацией.
Кроме того, в вышеупомянутую систему по настоящему изобретению можно включить другую информацию. Предпочтительно такая другая упомянутая здесь информация может представлять собой данные по исследованию транскриптом, липидом и/или метаболом. Более предпочтительно можно использовать транскриптомные данные. Такие транскриптомные данные могут представлять, как правило, транскриптомные данные, полученные от субъекта или группы субъектов после введения лекарственного средства, или транскриптомные данные, указывающие на различие между двумя группами субъектов или субъектом или группой субъектов до и после лечения с помощью лекарственного средства. Более предпочтительно использовать эту другую информацию, и предпочтительно, транскриптомные данные, для моделирования наборов данных в базе данных. Наиболее предпочтительно, наборы данных из базы данных удаляются, если не найдено совпадение между идентификатором молекулы в наборе данных и идентификатором молекулы в другой информации, предпочтительно, в транскриптомных данных. Обычно транскриптомные данные содержат информацию об уровне экспрессии транскриптов для затравочных молекул, регуляторов или компонентов по взаимодействию, содержащихся в сетевой карте. Данные об уровнях экспрессии могут представлять собой параметры, отражающие абсолютные уровни или относительные изменения. Более того, могут использоваться любые математические значения, полученные с использованием таких параметров, например, логарифмы. Более подробно такие методики по включению транскриптомных данных в карту сети представлены ниже, и особенно в сопутствующих примерах.
Более предпочтительно, карту сети, которую можно генерировать и использовать с помощью системы по изобретению, можно создать по схеме, включающей следующие стадии.
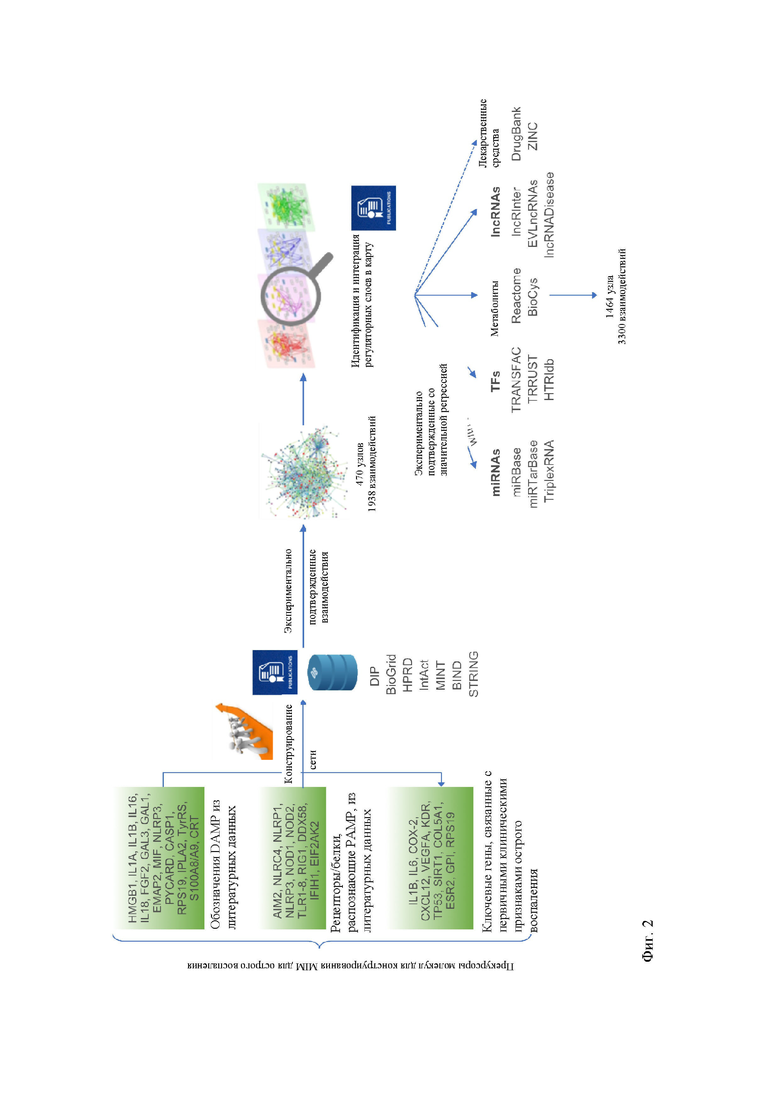
Более подробно, на первой стадии, затравочные молекулы, которые принимают участие в моделируемых молекулярных взаимодействиях, идентифицируют в ходе анализа научной литературы по образ-распознающим рецепторам DAMP, РАМР и по клиническим признакам острого воспаления. Наборы данных для этих затравочных молекул представлены в базе данных (см. фиг. 2).
Затем карту сети конструировали при дальнейшем анализе базы данных и литературы с целью идентификации компонентов по взаимодействию и/или регуляторов затравочных молекул. Обычно поиск можно выполнять в следующих базах данных: DIP, BioGrid, HPRD, IntAct, MINT, BIND и/или STRING. Взаимодействие может быть подтверждено на основе экспериментальных данных, имеющихся в литературе. Полученную взаимосвязь затравочных молекул друг с другом, их компонентов и регуляторов по взаимодействию включают в карту сети. Затравочные молекулы, компоненты и регуляторы по взаимодействию также сгруппированы в функциональные секции биологической функции при воспалении на основе информации, содержащейся в наборе данных для каждой затравочной молекулы (см. фиг. 2).
Наконец, дополнительную информацию можно интегрировать в карту и использовать для подтверждения регуляторных взаимодействий, полученную карту сети можно визуализировать и исследовать, как подробно описано выше (фиг. 2).
Преимущество настоящего изобретения заключается в том, что неожиданно было установлено, что систему для моделирования молекулярных взаимодействий можно предоставить на основе сетевой карты молекулярных взаимодействий между предварительно заданными затравочными молекулами и соответствующими компонентами и регуляторами по взаимодействию. Такая сетевая карта обеспечивает основу для динамического системного анализа заболеваний. Молекулярные взаимодействия на карте представляют собой физические или причинно-следственные связи между объектами, которые можно преобразовать в математические уравнения для анализа динамики системы в течение времени. Формализм логического моделирования (Boolean) можно использовать для моделирования динамического системного поведения сети для различных фенотипов заболеваний в различные моменты времени. Для логических моделей не требуются подробные количественные кинетические параметры, что делает их пригодными для динамического анализа больших регуляторных сетей. Логические функции можно обучить и/или откалибровать с использованием экспериментальных данных, чтобы придать им специфичность согласно контексту/типу клетки/фенотипу заболевания. После калибровки модели с использованием экспериментальных данных можно проводить эксперименты in silico для имитации известного поведения системы в зависимости от различных стимулов или исходных условий. Кроме того, модель будет подвергаться различным возмущениям, при этом изменение логических состояний узла(ов) будет наблюдаться в контексте фенотипических результатов (например, устранение воспаления). Такие эксперименты в связи с возмущениями можно использовать для выявления наиболее существенных взаимодействий, которые могут представлять интерес в качестве мишеней для терапии.
Далее более подробно описаны предпочтительные варианты осуществления системы по настоящему изобретению. Приведенные выше объяснения и определения терминов следует применять с соответствующими поправками mutatis mutandis, если не указано иное.
В предпочтительном варианте системы по изобретению указанное воспаление включает устранение воспаления.
В еще одном предпочтительном варианте указанную молекулу идентифицируют из ассоциированных с повреждением образ-распознающих молекул (DAMP) или из патоген-ассоциированных образ-распознающих молекул (РАМР). Более предпочтительно, упомянутую DAMP и/или упомянутую РАМР получают в ходе оценки публикаций в общедоступных научных базах данных с использованием автоматического алгоритма оценки, который идентифицирует молекулы, предположительно принимающие участие в воспалительном процессе.
В предпочтительном варианте осуществления системы по изобретению биологическая функция молекулы в воспалительном процессе выбрана из группы, состоящей из дегрануляции тучных клеток, дифференциации макрофагов, дифференциации миелоидных клеток, дифференциации лимфоцитов, дифференциации иммунных клеток, регуляции гемопоэза, стимуляции врожденного иммунного ответа, сигнальных путей образ-распознающих рецепторов, продуцирования цитокинов, регуляции приобретенного иммунного ответа, опосредованного Т-клетками иммунного ответа, селекции Т-клеток, регуляции пролиферации В-клеток, регуляции секреции иммуноглобулинов, регуляции пролиферации Т-клеток, регуляции пролиферации лимфоцитов, разрешения воспаления, регуляции экспрессии генов, модификации белков, развития кровеносных сосудов, иммунного ответа, гемопоэза, развития нейронов и транспорта белка.
В предпочтительном варианте осуществления системы по изобретению, указанный алгоритм на основе компьютерной программы, реализованный в блоке обработки, который генерирует карту сети на основе множества наборов данных в базе данных и который позволяет идентифицировать узлы в карте сети, содержит правила для приоритизации генов, определения степени узла, определения центральности по посредничеству, идентификации мотива, в частности для идентификации петель обратной связи или прямой связи и/или определения ассоциации с воспалением.
В предпочтительном варианте осуществления системы по изобретению транскриптомные данные, полученные от субъекта после введения лекарственного средства, также представлены в базе данных. Более предпочтительно, транскриптомные данные используются для моделирования наборов данных в базе данных. Наиболее предпочтительно, наборы данных удаляются из базы данных, если не найдено совпадение между идентификатором молекулы в наборе данных и идентификатором молекулы в транскриптомных данных.
В предпочтительном варианте осуществления указанной выше системы по изобретению указанным лекарственным средством является многокомпонентное лекарственное средство. Более предпочтительно, указанное многокомпонентное лекарственное средство представляет собой траумель (Traumeel).
В предпочтительном варианте осуществления системы по изобретению блок визуализации содержит алгоритм на основе компьютерной программы, который графически размещает узлы с высокой степенью взаимосвязи друг с другом и/или который идентифицирует для графического отображения только узлы, которые связаны внутримодулярно.
В настоящем изобретении предлагается также способ моделирования молекулярных взаимодействий, принимающих участие в воспалительном процессе у субъекта, который включает:
(I) обеспечение блока обработки данных, включающего:
(а) базу данных, включающую множество наборов данных, каждый из которых включает:
(i) по меньшей мере один идентификатор молекулы, для которой предполагается участие в патологическом процессе,
(ii) данные по молекулярным взаимодействиям указанной молекулы с одной или более других молекул, и
(iii) по меньшей мере одну характеристику данных, выделенную для набора данных, где каждый набор данных связан по меньшей мере в одном отношении с другим набором данных в базе данных, основанной на молекулярных взаимодействиях,
где наборы данных сгруппированы в секции данных, включающие наборы данных, содержащих идентичные характеристики данных; и
где характеристики данных указывают на биологическую функцию молекулы воспалительного процесса;
и
(б) алгоритм на основе компьютерной программы, включенный в блок обработки данных, который генерирует карту сети, основанную на множестве наборов данных в базе данных и который позволяет идентифицировать узлы в карте сети на основании предварительно определенных параметров; и
(II) генерирование карты сети, основанной на множестве наборов данных в базе данных, и идентификация узлов в карте сети, основанной на предварительно определенных параметрах в ходе выполнения алгоритма на основе компьютерной программы, реализованного в блоке обработки, и
(III) определение молекулярных взаимодействий, принимающих участие в воспалительном процессе, в идентифицированных узлах с использованием блока визуализации, с помощью которого моделируются молекулярные взаимодействия, принимающие участие в воспалительном процессе у субъекта.
В предпочтительном варианте способа по изобретению, указанное воспаление включает устранение воспаления.
В другом предпочтительном варианте осуществления способа по изобретению указанную молекулу идентифицируют из ассоциированных с повреждением образ-распознающих молекул (DAMP) или из патоген-ассоциированных образ-распознающих молекул (РАМР). Более предпочтительно, упомянутый DAMP и/или упомянутый РАМР получают в ходе оценки публикаций в общедоступных научных базах данных с использованием автоматического алгоритма оценки, который идентифицирует молекулы, предположительно принимающие участие в воспалительном процессе.
В предпочтительном варианте осуществления способа по изобретению указанная биологическая функция молекулы в воспалительном процессе выбрана из группы, состоящей из дегрануляции тучных клеток, дифференциации макрофагов, дифференциации миелоидных клеток, дифференциации лимфоцитов, дифференциации иммунных клеток, регуляции гемопоэза, стимуляции врожденного иммунного ответа, сигнальных путей образ-распознающих рецепторов, продуцирования цитокинов, регуляции приобретенного иммунного ответа, опосредованного Т-клетками иммунного ответа, селекции Т-клеток, регуляции пролиферации В-клеток, регуляции секреции иммуноглобулинов, регуляции пролиферации Т-клеток, регуляции пролиферации лимфоцитов, разрешения воспаления, регуляции экспрессии генов, модификации белков, развития кровеносных сосудов, иммунного ответа, гемопоэза, развития нейронов и транспорта белка.
В предпочтительном варианте осуществления способа по изобретению указанное создание карты сети на основе множества наборов данных в базе данных и идентификация узлов в карте сети включает приоритизацию генов, определение степени узла, определение центральности по посредничеству, идентификацию мотива, прежде всего, идентификацию петель обратной связи или прямой связи и/или определение ассоциации с воспалением.
В предпочтительном варианте способа по изобретению транскриптомные данные, полученные от субъекта после введения лекарственного средства, также представлены в базе данных. Более предпочтительно, транскриптомные данные используются для моделирования наборов данных в базе данных. Наиболее предпочтительно, наборы данных удаляются из базы данных, если не найдено совпадение между идентификатором молекулы в наборе данных и идентификатором молекулы в транскриптомных данных.
В предпочтительном варианте осуществления способа по изобретению указанным лекарственным средством является многокомпонентное лекарственное средство. Более предпочтительно, указанное многокомпонентное лекарственное средство представляет собой траумель (Traumeel).
В предпочтительном варианте осуществления способа по изобретению блок визуализации содержит алгоритм на основе компьютерной программы, который графически размещает узлы с высокой степенью связи друг с другом и/или который идентифицирует для графического отображения только узлы, которые связаны внутримодулярно.
В настоящем изобретении предлагается также применение системы по изобретению для моделирования молекулярных взаимодействий, принимающих участие в воспалительном процессе у субъекта. Более предпочтительно, указанное моделирование молекулярных взаимодействий, принимающих участие в воспалительном процессе у субъекта, используют для определения действия лекарственного средства.
Все процитированные в настоящем описании публикации включены в него в полном объеме, также как конкретно упомянутое содержание их раскрытия.
Фигуры
Фиг. 1. Пользовательский интерфейс инструмента, который разработан авторами настоящего изобретения для хранения данных и интеграции функций, которые позволяют автоматизировать обработку, например, для экспорта данных в систему карты молекулярных взаимодействий (MIM).
Фиг. 2. Последовательность действий для конструирования комплексной MIM по острому воспалению. Сначала были идентифицированы затравочные молекулы (левая панель на фигуре). Затем были извлечены экспериментально подтвержденные взаимодействующие молекулярные компоненты из нескольких баз данных и литературных источников (в центре). Наконец, были добавлены различные регуляторные уровни (miRNA, TF, IncRNA) из баз данных уровня техники. Также были включены метаболиты, ассоциированные с разрешением воспаления. Могут быть интегрированы дополнительные регуляторные слои, такие как молекулы лекарственного средства (показаны справа).
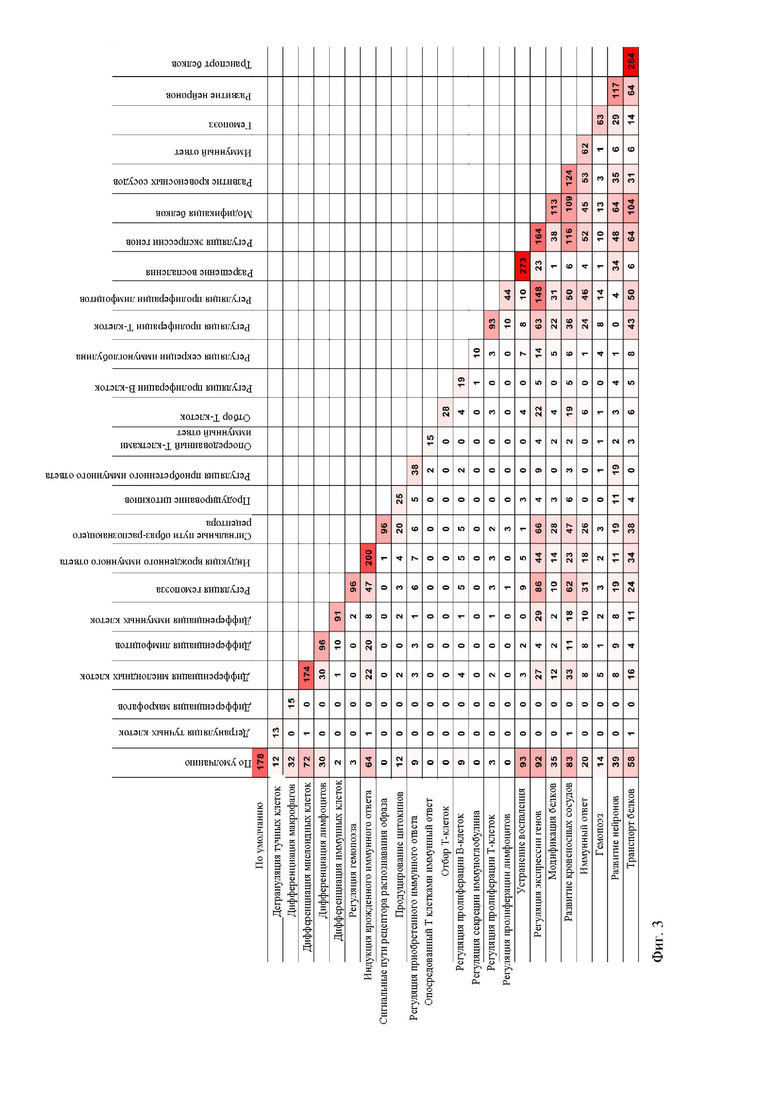
Фиг. 3. Взаимосвязанность сети отображается как количество взаимодействий между двумя модулями.
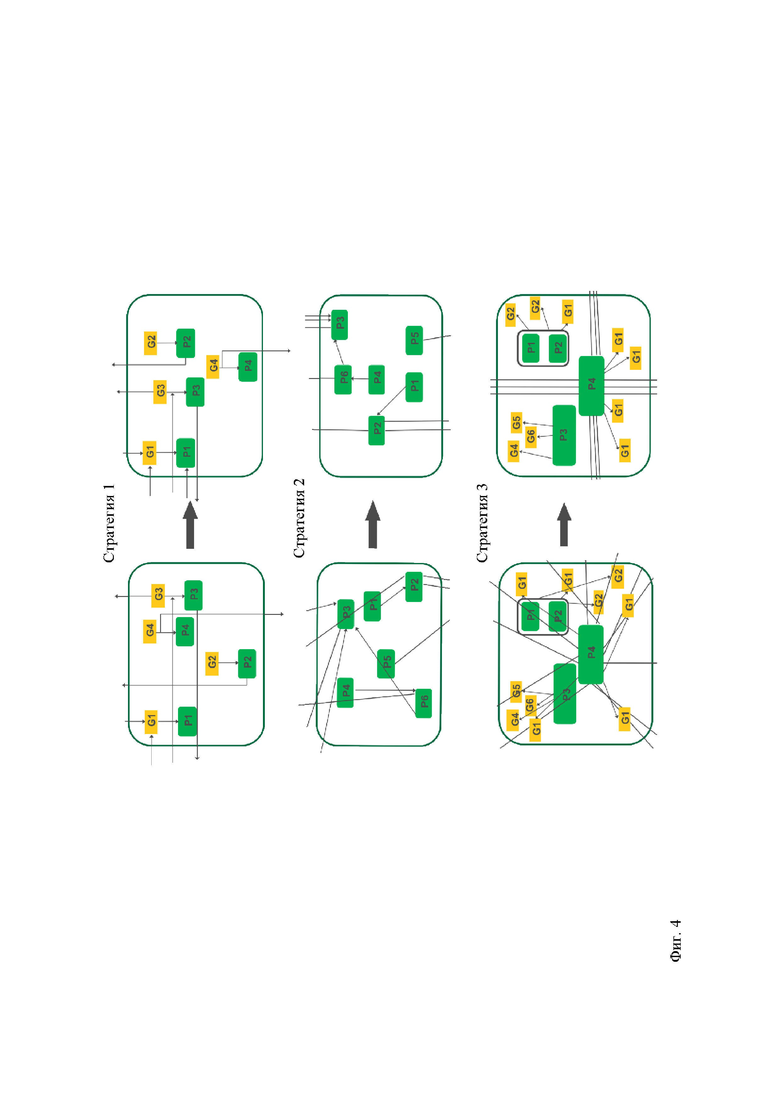
Фиг. 4. Различные этапы для расположения сетевых компонентов и взаимодействия между ними.
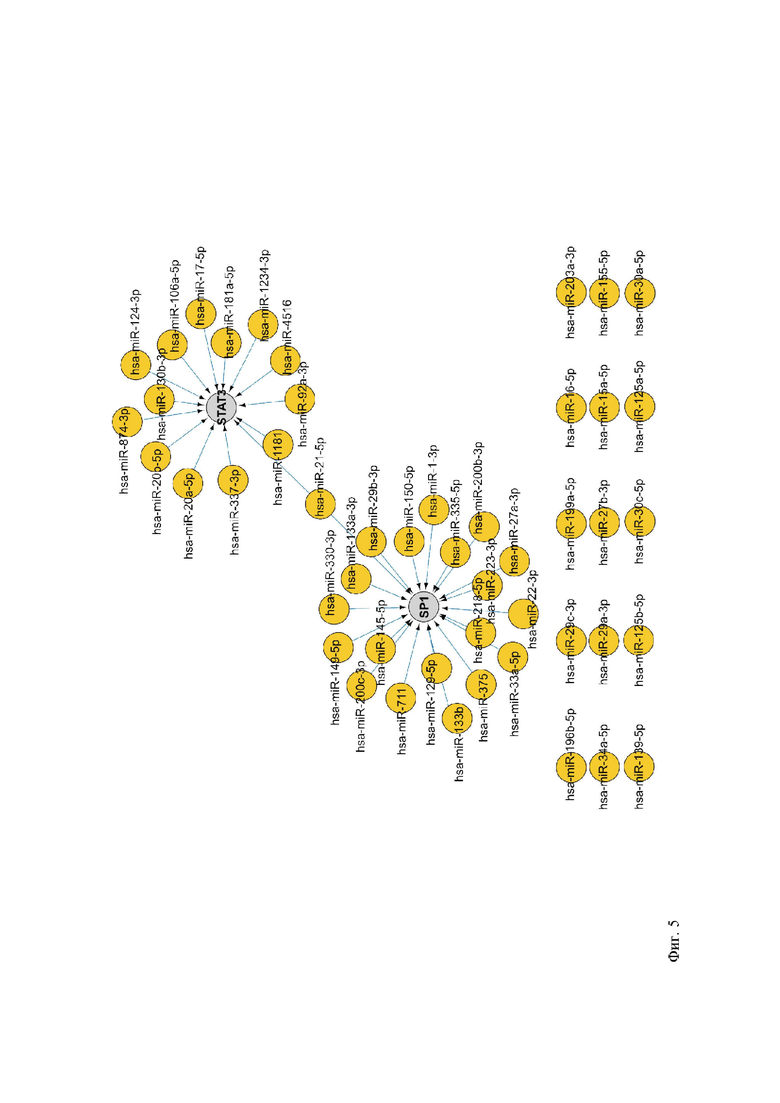
Фиг. 5. Обычное регуляторное ядро среди всех первичных клинических признаков острого воспаления MIM. TF (формы представления, факторы транскрипции) показаны в виде серых узлов, а микро РНК - в виде оранжевого узла.
Фиг. 6. Общие молекулы среди 5 первичных клинических признаков острого воспаления. Доля бурсита составляет 86% узлов с тендинитом, в то время как 40% всех узлов являются общими с теносиновитом.
Фиг. 7. Нанесение на карту 1000 наиболее активированных генов (top 1000 up-regulated genes) с онтологиями генов/фенотипов. Тысяча (1000) наиболее активированных генов транскриптома мыши были сопоставлены с онтологиями генов, связанных с воспалением, в зависимости от времени. На графиках указано количество генов, ассоциированных с конкретным процессом в перечне top 1000, в виде процентов от всего GO-набора (GeneOntology).
Примеры
Следующие примеры представлены только для иллюстрации настоящего изобретения. Они ни в коем случае не должны рассматриваться как ограничение объема заявленных притязаний.
Пример 1
Создание сетевой карты молекулярных взаимодействий при воспалении (карта молекулярных взаимодействий, MIM)
Идентификация затравочных молекул для конструирования MIM
1. Конструирование MIM является высоко структурированным рабочим процессом, который начинается с нескольких затравочных молекул, которые в дальнейшем дополняются экспериментально подтвержденными молекулярными компонентами и несколькими уровнями регуляторной информации. Затравочные молекулы для конструирования MIM были идентифицированы в три этапа:
2. Скрининг ассоциированных с повреждением образ-распознающих молекул (DAMP)
3. Скрининг патоген-ассоциированных образ-распознающих молекул (РАМР)
4. Анализ выбранных клинических показаний острого воспалительного процесса.
DAMP и РАМР идентифицировали в ходе поиска вручную научной литературы с использованием более 50 оригинальных исследовательских работ и обзорных статей. В основном обращали внимание на DAMP, которые часто упоминаются в литературе по острым воспалениям из-за повреждения мышц/соединительных тканей и физических травм (Таблица 1).


Была изучена литература по исследованию РАМР, относящаяся к острому воспалению, вызванному инфекцией различными патогенами. Инородные частицы взаимодействуют с рецепторами, при этом запускаются иммунные ответы, и поэтому ассоциированные рецепторы использовали в качестве затравочных молекул для построения MIM. Перечень РАМР и их распознающих рецепторов представлен в Таблице 2 ниже.
Чтобы MIM оставалась близкой к клинической значимости, также был проведен анализ литературы (> 50 публикаций и > 15 обзорных статей) по молекулярным событиям, связанным с различными острыми воспалительными клиническими показаниями. Был проанализирован список 29 первичных клинических показаний острого воспаления, предоставленный HEEL GmbH, была проведена проверка общедоступной литературы (PubMed, OMIM) и базы данных генов, ассоциированных с заболеванием (DisGeNET, DISEASE, болезнь KEGG) для выявления генов, связанных с клиническим показанием. Большинство этих баз данных основаны на алгоритмах интеллектуального анализа текста для прогнозирования взаимосвязи между биологическими объектами, что во многих случаях приводит к ложноположительной информации. Связь ключевых генов с клиническими показаниями была анализировали вручную при скрининге соответствующей публикации. Ключевые гены, связанные с каждым из основных клинических признаков острого воспаления, сведены в Таблицу 3.
Наконец, был создан уникальный набор генов из Таблиц 1-3 в качестве затравочных молекул, которые были использованы для создания комплексной MIM. Всего были использованы 53 затравочных молекулы, которые были использованы для создания MIM.
Создание MIM из затравочных молекул (неориентированный граф) Для всех затравочных молекул была вручную собрана информация о молекулярных взаимодействиях в области острого воспаления из литературных данных, а также из нескольких автоматизированных инструментов, таких как приложение BisoGenet, доступное на Cytoscape4.0, которое объединяет большое количество баз данных.
Прежде всего, была собрана следующая информация:
- исходные молекулы и молекулы-мишени,
- возможные модификаторы (например, ферменты),
- тип модификации (например, катализ, фосфорилирование),
- состояние молекулы (например, фосфорилированная),
- тип молекул (белки, микроРНК, комплексы, ДНК, простые молекулы),
- тип взаимодействия (положительный, отрицательный),
- ссылки на PubMed-ID.
Чтобы структурировать информацию в одном месте и объединить данные, полученные из других источников (например, база данных STRNG), был создан инструмент, который позволяет легко хранить, обрабатывать и изменять данные и дает возможность реализовать новую функцию для управления данными в связи с предстоящими задачами. Инструмент был создан на фирме С # Microsoft.NET Framework с использованием Microsoft Visual Studio Community 2017 версии 15.9.0. На фигуре 1 показан пользовательский интерфейс в текущем состоянии разработки (декабрь 2018 г.).
С этой целью были получены все затравочные молекулы и извлечена информация о первых экспериментально подтвержденных молекулярных мишенях, а также прямые взаимодействия между ними, если они были предоставлены. В основном рассматривались экспериментально подтвержденные молекулярные мишени из баз данных DIP, BioGrid, HPRD, IntAct, MINT, BIND и String. Кроме того, было подключено несколько экспериментально подтвержденных регуляторных слоев, которые включают MHKpoRNAs из mirBase, MHKpoRTarBase, TriplexRNA, факторы транскрипции из баз данных TRNSFAC, TRRUST и HTRIdb; lnc РНК из IncRInter, EVLncRNAs, IncRNADisease. Уже очевидно, что метаболизм жирных кислот играет важную роль в биосинтезе специализированных про-разрешающих молекул (включая резолвины, протектины и марезины). Также была собрана информация о биосинтезе этих медиаторов разрешения воспаления из базы данных Reactome и опубликованной литературы, и они были включены в MIM вместе со всеми ассоциированными ферментами. Общий процесс представлен на фигуре 2. В настоящее время версия сети cytoscape включает 1464 узла и 3300 взаимодействий.
Аннотации и обогащение MIM
Каждый ген в MIM был обогащен своими конкретными идентификаторами UNIPROT-, HGNC-, RefSeq-, Ensemble-, PubMed- и NCBI-ID, а также общепринятыми псевдонимами и полным названием кодирующего белка. Эта информация была собрана в виде единого файла, загруженного 14 ноября 2018 г. из базы данных HGNC. В случае простых молекул, CHEBI-ID был извлечен для каждой молекулы отдельно из базы данных CHEBI при сборе данных о взаимодействии из литературы.
Модульная организация MIM на основе процессов, связанных с острым воспалением
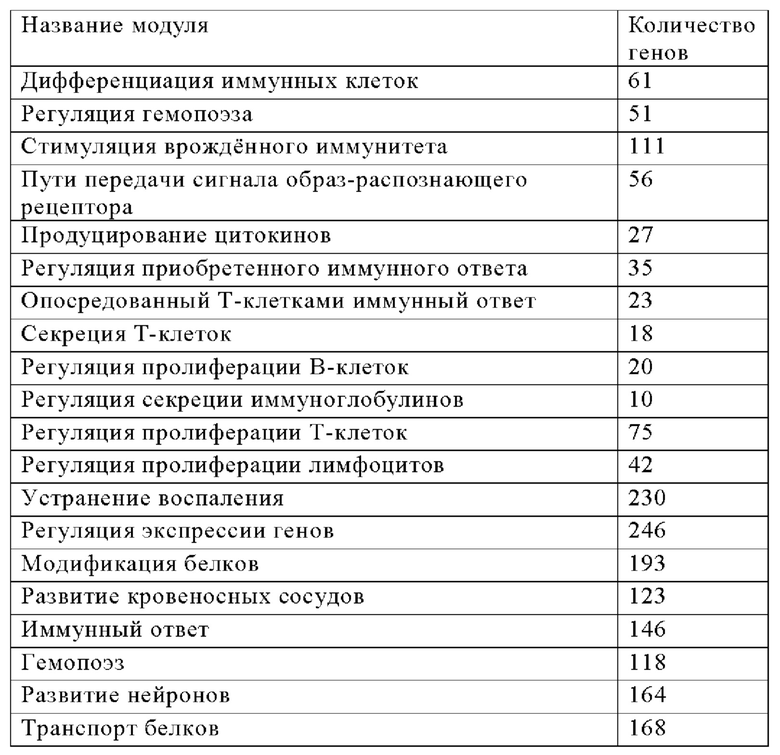
Для упрощения навигации и визуализации MIM, карта MIM была разделена на различные функциональные модули при присвоении молекулам терминов генной онтологии, ориентированным на острое воспаление. Для этого использовали плагин CytoScape ClueGO, который использует базу данных GeneOntology (GO) для структурирования списка генов в модули GO-Terms. Результаты можно скорректировать вручную, задав параметры, например, диапазон уровней дерева термина в GO-Hierarchy, р-значение для ассоциации или количество генов в модулях. Чтобы сфокусировать внимание на процессы, связанные с иммунологией, база данных GO «immmunological process)) была использована в первом прогоне модуляризации, и 230 из всех 1464 (фиг. 2) генов были назначены 116 терминам GO, которые были вручную объединены в 17 модулей. Во втором прогоне использовали более общую базу данных «Biological process)), и 778 из оставшихся 1234 генов были присвоены 79 терминам GO, которые снова были объединены в семь модулей.
Модуль устранения воспаления
Основная цель разработки и построения MIM заключалась в том, чтобы понять процессы острого воспаления и устранения воспаления. Поскольку процесс устранения воспаления не включен в базу данных GeneOntology, гены были вручную назначены этому модулю на основе информации, доступной в литературе. Было обнаружено большое количество специализированных про-разрешающих медиаторов (SPM), для которых информация о биосинтезе и сигнальных путях была извлечена из литературы (PubMed) и базы данных путей (Reactome). Неферментативным медиаторам были присвоены названия по систематической номенклатуре, которая объединяет синонимы, используемые в различных публикациях. Номенклатура основана в основном на названии ChEBI (https://www.ebi.ac.uk/chebi/). Ферментам и белкам были присвоены названия, соответствующие названиями генов UniProt (https://www.uniprot.org/). Величина/направление взаимодействия определяли как положительное, отрицательное или нейтральное (с дополнительными уточнениями, при необходимости, например, «нейтральное (транслокация от нейтрофилов к тромбоцитам)»), а тип взаимодействия назван в соответствии с терминологией (например, «катализ», «инактивация», «фосфорилирование» и др.). Если взаимодействие представляет собой стадию биосинтеза или инактивации, продукт реакции далее характеризуется либо как метаболит, либо как медиатор. Кроме того, при наличии добавляется информация о типах клеток, связанных с взаимодействием. Наконец, на каждое взаимодействие приводится ссылка на публикации PMID, использованные для получения информации о нем. На данный момент этот модуль содержит 256 взаимодействий, включая (не ограничиваясь ими) пути биосинтеза протектинов, марезинов, резолвинов, липоксинов, соответствующих предшественников, соответствующих путей инактивации и ассоциированных рецепторов. В Таблице 4 указаны все 25 модулей и их общее количество генов, в которых ген может встречаться в нескольких модулях.

Затем в карту MIM в качестве модуля медиаторов воспаления были также включены пути биосинтеза различных медиаторов воспаления (лейкотриены, эоксины, простагландины, тромбоксаны, гепоксилины и триоксилины) наряду с их предшественниками и соответствующими путями инактивации
Визуализация сети
Для визуализации молекулярных взаимодействий в карте молекулярных взаимодействий MIM необходимо было сконструировать приложения, в которых можно создавать и выводить на экран эту информацию в системах графического описания системной биологии (SBGN). Более того, эти приложения позволяют визуализировать и анализировать экспериментальные данные в карте. Из-за большого размера MIM создание вручную практически невозможно или требует значительного количества времени и рабочей силы. Чтобы преодолеть эти недостатки, была разработана функция автоматического экспорта из набора данных в формат файла языка разметки системной биологии (SBML), который описывает MIM в стиле SBGN и его можно прочитать в приложении CellDesigner™, которое использовали в качестве инструмента для отображения и расположения карты, а также для интеграции транскриптомных данных во временной последовательности. Функция автоматически помещает все молекулы в соответствующие модули на карте и придает форму и цвет узлу в зависимости от типа молекулы, а также приводит схему взаимодействия. Взаимосвязь модулей визуализирована на фигуре 3 ниже, где указано число взаимодействий между двумя модулями.
Макет карты MIM для упрощения навигации
Карты молекулярного взаимодействия (MIM) являются важным источником молекулярных механизмов заболеваний, которые можно использовать для формулировки новых гипотез, которые впоследствии могут быть проверены экспериментально. Такие карты характеризуются большим размером (т.е. содержат большое количество компонентов) и сложностью (т.е. содержат трудные для понимания повторяющиеся структуры), что усложняет их визуализацию и исследование. Улучшенная визуализация может способствовать пониманию основных механизмов и позволит эффективно обмениваться данными и использовать их в биомедицинских исследованиях. Хотя инструменты построения и визуализации карт, такие как CellDesigner и Cytoscape, предоставляют встроенные варианты расположения, но в случае больших и модульных MIM они не могут обеспечить эффективную визуализацию. Алгоритмы автоматического расположения достаточно эффективно работают только для небольших карт. Для визуализации больших MIM требуется проведение множества операций вручную. В связи с этим были разработаны постадийные процедуры для организации сетевых компонентов и взаимодействий для улучшенной визуализации.
Стадия 1
Компоненты размещали в модуль на основе их взаимодействия с другими моделями (то есть межмодульные соединения) таким образом, чтобы взаимодействующие стрелки не пересекали друг друга. Например, на фиг. 4а слева показано соединение генов (G1,…,4) и белков (Р1,…,4) с другими модулями на карте.
В данном случае:
- G1, G3 и Р3 взаимодействуют с левой частью сети
- G1, G2, Р2 взаимодействуют с верхней частью сети
- G1 и G4 взаимодействуют с нижней частью сети.
В данном контексте, на основе расположения межмодульных взаимодействий, компоненты расположены либо в левой, верхней либо в нижней части модуля.
Стадия 2
Компоненты сети расположены на основании их взаимодействий в модуле, а также их взаимодействия с другими модулями карты, как показано на фиг. 4б.
Стадия 3
На этом этапе были решены другие проблемы с удобочитаемостью для таких компонентов, как белки и комплексы, которые имеют множество меж- и внутримодульных связей (фиг. 4в).
Масштабируемое изображение MIM в браузере
Задача заключалась в том, чтобы включить MIM в браузер для упрощения связи с сообществом, имеющим отношение к острому воспалению. Сначала была изучена литература по алгоритмам и методам отображения MIM в виде интерактивного изображения. Методы, основанные на сайтах OpenLayer и Google Maps, были выбраны для переноса MIM в браузер для упрощения визуализации. В частности, MIM была разделена на три уровня. Базовые уровни представляют собой модули на верхнем уровне, подмодули и виды на среднем уровне и виды и взаимодействия на нижнем уровне. Для уменьшения вычислительных затрат и требований к локальному интернету было выполнено мозаичное разбиение изображения. Мозаичное разбиение представляет собой метод, позволяющий разрезать изображения на матрицу меньших изображений одного и того же размера и сохранять их в определенной структуре папок. При этом можно загрузить и отобразить только просматриваемые в данный момент части MIM. Были использованы сценарии Python для мозаичного и многослойного размещения, чтобы проверить, является ли пригодным экспорт нашей карты CellDesigner для процесса обработки информации MIM перед отправкой конечному пользователю. Для визуализации карты использовали платформу MINERVA [https://minerva.pages.uni.lu]. Был установлен локальный экземпляр MINERVA и протестирован с картой MIM на предмет различных проблем с безопасностью и надежностью.
MINERVA предоставляет возможность использовать OpenLayer в качестве базовой библиотеки визуализации для повышения безопасности данных по сравнению с GoogleMaps. Процесс мозаичного разбиения и наложения полностью автоматизирован, также поддерживаются базовые операции мыши для масштабирования и перемещения окна MIM. Кроме того, пользователь может выбирать узлы для просмотра аннотаций и одновременно подключаться к различным базам данных лекарственных средств и химических реагентов непосредственно из интерфейса MINERVA для дальнейшего анализа сети.
Создание MIM в виде ориентированного графа
Обеспечение направления (например, активация, ингибирование) к краям, соединяющим различные узлы в сети, является важным шагом для анализа и прогнозирования биомаркеров и терапевтических кандидатов. Это один из самых важных шагов в конструировании надежной карты заболеваний, требующий проведения множества операций вручную. Были проанализированы все ассоциированные публикации, в которых основное внимание направлено на взаимодействие между биологическими компонентами, чтобы найти тип регуляции и соответствующим образом описывать реакции. Использовали также ресурсы системной биологии, такие как базы данных BioModel, для назначения нормативных указаний подключенным компонентам с использованием внутренних сценариев. Многие базы данных используют методы интеллектуального анализа текста, чтобы даже выделить экспериментально подтвержденные взаимодействия с ложноположительной информацией. В то время при генерировании MIM заболеваний проводили анализ для определения типа регуляции (например, активация, подавление, активированный комплекс и т.д.). Тем не менее, этот анализ стоит проделать для надлежащего тестирования MIM, а также для последующего анализа, связанного с идентификацией мотивов регуляции и установлением приоритетов сетевых компонентов. На данный момент около 70% всех взаимодействий в сети являются направленными.
Идентификация общих регуляторов из выбранных первичных клинических показаний острого воспаления
Были выявлены некоторые из первичных клинических признаков острого воспаления, информация о ключевых генах которых была доступна в литературе.
Для этих клинических показаний необходимо найти общие молекулярные регуляторы. Для решения этой задачи сначала была сконструирована MIM, специфичная для клинических показаний, с использованием рабочего процесса, описанного на фиг. 2.
Для регуляторного слоя микроРНК мы использовали сайт микроRTarBase release 6.1 и рассматривали только те микроРНК, которые, как экспериментально показано, обладают высокой подавляющей эффективностью в отношении генов-мишеней. Перечень ключевых генов, число узлов, взаимодействий, число известных факторов транскрипции и число микроРНК, строго регулирующих гены-мишени в сетях, показаны в Таблице 5.
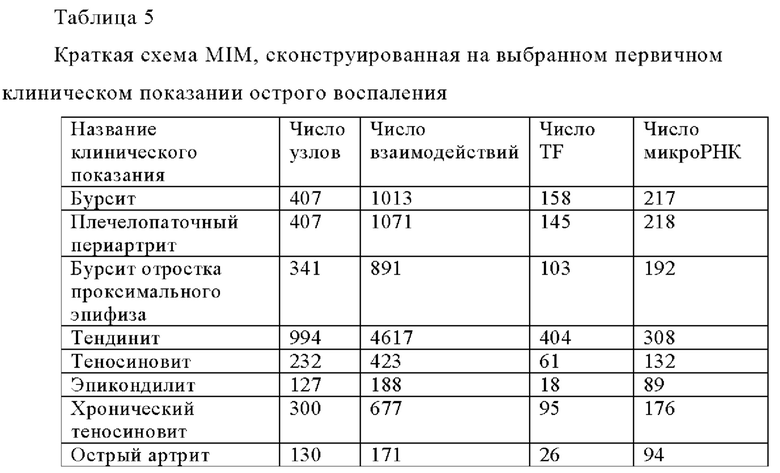
Чтобы идентифицировать общие гены, TF и микроРНК, общие для всех исследуемых здесь основных клинических показаний, был использован сетевой инструмент слияния Cytoscape, чтобы выявить взаимодействия между сетями, специфичными для клинических показаний. Факторы транскрипции STAT3 и SP1 были обнаружены наряду с 48 экспериментально подтвержденными микроРНК в качестве общих регуляторов по всем 8 первичным клиническим показаниям острого воспаления, для которых была сконструирован MIM (фиг. 4).
В случае бурсита, тендинита, тендосиновита, эпикондилита и острого артрита было выявлено 54 общих кандидата (фиг. 5), включая 4 фактора транскрипции (EGR1, STAT3, SP1 и JUN) и 50 микроРНК. Ранее сообщалось о роли STAT3 за счет передачи сигналов, управляемой интерлейкином IL-6, в остановке воспалительного накопления нейтрофилов, что является важной контрольной точкой в разрешении воспаления (PMID: 18641358). Среди общих микроРНК мы обнаружили miR-155, которая была идентифицирована как центральный регулятор иммунной системы (PMID: 19596814). Среди прочего, уже известна роль miR-21 и miR-203a в разрешении острого воспаления (PMID: 20956612).
Пример 2
Интеграция транскриптомных данных траумеля с использованием модели ранозаживления у мышей в карту MIM и исследования индуцированного траумелем фенотипа
Интеграция транскриптомных данных траумеля с использованием модели ранозаживления у мышей в карту MIM
Данные последовательностей РНК RNA-seq, предоставленные в базе HEEL, охватывают 55419 транскриптов, также частично кодирующих изомеры генов. Уровни экспрессии генов определяли количественно при измерении значений RPKM (считываний на килобазу на миллион прочтений) для каждого транскрипта. Значения RPKM были измерены для 6-7 образцов (n=6/7), в 8 временных точках (0 ч - 192 ч) и для 4 условий (нелеченые, леченые физиологическим раствором, только инъекцией траумеля, инъекцией траумеля плюс мазь). Было установлено, что на сайте RNA-seq значения RPKM были представлены для каждого из транскриптов, ассоциированных с конкретным геном. Затем в процессе идентификации и анализа были выявлены дифференциально экспрессируемые гены и их связи с процессами более высокого уровня при картировании терминов GO. До сих пор ассоциация терминов GO на уровне транскриптов генов в значительной степени отсутствует, и, как правило, на карту наносятся одни и те же термины GO для всех транскриптов генов. Чтобы быстро оценить дифференциально экспрессируемые гены по данным RNA-seq в различных условиях и отобразить их на карте MIM, значения RPKM были добавлены для всех изомеров гена для каждой репликационной копии отдельно с последующим расчетом их среднего значения в различных условиях. Затем рассчитывали логарифмическое log(2)-кратное изменение каждого гена при сравнении различных условий (нелеченые мыши по сравнению с инъекцией только траумеля; инъекция физиологического раствора по сравнению с инъекцией только траумеля; нелеченые мыши по сравнению с нанесением геля траумель; и инъекция физиологического раствора по сравнению с нанесением геля траумеля). Кроме того, данные по логарифмическому log(2)-кратному изменению были интегрированы в карту MIM для каждой временной точки. Из версии MIM в cytoscape были извлечены дифференциально выраженные сетевые компоненты (>1,5 log(2)-кратное изменение и р-значение <0,05) в условиях эксперимента на нелеченых мышах по сравнению с инъекцией траумеля через 12 часов. Изменение характера экспрессии этой дифференциально экспрессируемой сети исследовали в более поздний момент времени. Через 120 часов для множества узлов наблюдался одинаковый профиль экспрессии при сравнении двух вышеупомянутых условий эксперимента. Через 192 часов ни для одного из узлов профиль экспрессии на изменялся, что указывает на то, что в этот момент времени уже произошло устранение воспаления.
Оценка влияния лечения траумелем на биологические процессы и фенотипы - на основе log(2)-кратного изменения экспрессии генов
В первой попытке определить, как траумель влияет на основные регуляторные процессы в модели ранозаживления у мышей, были идентифицированы наборы генов для выбранных биологических процессов и фенотипов с использованием сайта Gene Ontology Consortium (GO, http://www.geneontology.org) и базы данных Mouse Phenome (MP, https://phenome.jax.org). Затем были извлечены предварительно обработанные данные зависимости от времени (усреднение изоформ и образцов генов) для каждого набора генов GO/MP из транскриптомных данных и визуализированы на графике линейной зависимости.
Чтобы получить представление о поведении выбранных биологических процессов и фенотипов в целом (смещение акцента с отдельных молекул на сложные функции), средние значения экспрессии для каждой временной точки были рассчитаны и визуализированы в виде логарифмического log(2)-кратного изменения. Синяя линия показывает дифференциальную экспрессию всего набора генов, тогда как оранжевая, соответственно серая линия, представляет наборы генов только для физиологического раствора (S) и лечения только траумелем (Т). Физиологический раствор в этом контексте использовали в качестве эталона из-за предположения, что уже сама процедура инъекции, а также растворы-носители, например, физиологический раствор, может влиять на транскрипцию.
Оценка влияния лечения траумелем на биологические процессы и фенотипы - на основе 1000 наиболее активированных генов
Чтобы идентифицировать биологические процессы, на которые в наибольшей степени влияет лечение траумелем, были извлечены 1000 наиболее активированных генов для каждой временной точки (12 ч - 192 ч) у нелеченых (U) и леченых траумелем (Т) животных и отображали их на карте генных онтологий с использованием веб сервера для анализа обогащения набора генов EnrichR (http://amp.pharm.mssm.edu/Enrichr/). Таким образом, был сформирован и отправлен для дальнейшей оценки перечень 3624 GO-терминов. В качестве первой выборки мы извлекли все онтологии генов, связанных с Т-клетками, нейтрофилами, макрофагами и миграцией. Во-вторых, в качестве примера были выбраны два биологических процесса, в основном связанных с воспалением, а именно «миграция нейтрофилов» и «дегрануляция нейтрофилов».
На фиг. 7 указано количество генов, присутствующих в перечне 1000 наиболее активированных генов (top 1000) в каждый момент времени в процентах от всего набора генов, связанных с GO, где на ось абсцисс нанесены временные точки, а на ось ординат - количество генов, связанных с GO, в группе наиболее активированных генов (top 1000) в процентах от всего набора GO. В качестве примера: если GO-перечень содержит 480 генов и 48 из них присутствуют в перечне top 1000 в момент времени t=24 ч, значение у для этого момента времени будет составлять 10%.
Сравнение животных, леченых траумелем, с нелечеными животными (фиг. 7), позволяет сделать два основных вывода: во-первых, общая тенденция в отношении активированных генов, в процентах от GO-набора, не изменяется (то есть без нарушения физиологии). Во-вторых, траумель, по-видимому, сначала стимулирует экспрессию генов, участвующих в биологических функциях, связанных с воспалением (более эффективная инициация), а затем ингибирует вовлечение GO-набора в конце (более эффективное устранение). Такое направленное усиление процесса может быть объяснением способности траумеля вызывать устранение воспаления.
Список процитированной литературы
Dreyer et al. 2018, ВВА Molecular Basis of Disease 1864: 2315-2328;
Khan et al. 2018, Nature communications 8: 198
Netea et al. 2017, Nature Immunol. 18(8): 826-831
Sadeghi et al. 2016, PLOS one, DOI: 10.1371/journal.pone.0168760
Steffen et al. 2017, Journal of Biotechnology 261: 85-96
Wolkenhauer 2002, BioSystems 65: 1-18


Настоящее изобретение относится к биоинформатике. Раскрыта система для моделирования молекулярных взаимодействий, принимающих участие в воспалительном процессе у субъекта, включающая: (I) блок обработки данных, включающий: (а) базу данных, включающую множество наборов данных, каждый из которых включает: (i) по меньшей мере один идентификатор молекулы, для которой предполагается участие в патологическом процессе, (ii) данные по молекулярным взаимодействиям указанной молекулы с одной или более других молекул, и (iii) по меньшей мере одну характеристику данных, выделенную для набора данных, где каждый набор данных связан по меньшей мере в одном отношении с другим набором данных в базе данных, основанной на молекулярных взаимодействиях. При этом наборы данных сгруппированы в секции данных, включающие наборы данных, содержащих идентичные характеристики данных; и характеристики данных указывают на биологическую функцию молекулы в воспалительном процессе. Транскриптомные данные, полученные у субъекта после введения лекарственного средства, также предоставлены в базе данных, и эти транскриптомные данные используют для моделирования наборов данных в базе данных; и алгоритм на основе компьютерной программы, включенный в блок обработки данных, который генерирует карту сети, основанную на множестве наборов данных в базе данных и который позволяет идентифицировать узлы в карте сети на основании предварительно определенных параметров. Система также включает блок визуализации, который позволяет определять молекулярные взаимодействия, принимающие участие в воспалительном процессе, в идентифицированных узлах. Кроме того, представлен соответствующий способ моделирования молекулярных взаимодействий. Изобретение расширяет арсенал средств для моделирования молекулярных взаимодействий для оценки заболеваний и терапии заболеваний. 3 н. и 19 з.п. ф-лы, 7 ил., 5 табл., 2 пр.
1. Система для моделирования молекулярных взаимодействий, принимающих участие в воспалительном процессе у субъекта, включающая:
(I) блок обработки данных, включающий:
(а) базу данных, включающую множество наборов данных, каждый из которых включает:
(i) по меньшей мере один идентификатор молекулы, для которой предполагается участие в патологическом процессе,
(ii) данные по молекулярным взаимодействиям указанной молекулы с одной или более других молекул, и
(iii) по меньшей мере одну характеристику данных, выделенную для набора данных, где каждый набор данных связан по меньшей мере в одном отношении с другим набором данных в базе данных, основанной на молекулярных взаимодействиях;
где наборы данных сгруппированы в секции данных, включающие наборы данных, содержащих идентичные характеристики данных; и
где характеристики данных указывают на биологическую функцию молекулы в воспалительном процессе;
где транскриптомные данные, полученные у субъекта после введения лекарственного средства, также предоставлены в базе данных, и эти транскриптомные данные используют для моделирования наборов данных в базе данных;
и
(б) алгоритм на основе компьютерной программы, включенный в блок обработки данных, который генерирует карту сети, основанную на множестве наборов данных в базе данных и который позволяет идентифицировать узлы в карте сети на основании предварительно определенных параметров; и
(II) блок визуализации, который позволяет определять молекулярные взаимодействия, принимающие участие в воспалительном процессе, в идентифицированных узлах.
2. Система по п. 1, где указанный воспалительный процесс включает устранение воспаления.
3. Система по п. 1 или 2, где указанную молекулу идентифицируют из ассоциированных с повреждением образ-распознающих молекул (DAMP) или из патоген-ассоциированных образ-распознающих молекул (РАМР).
4. Система по п. 3, где указанный DAMP и/или указанный РАМР получают на основе оценки публикаций в общедоступных научных базах данных с использованием алгоритма автоматической оценки, который идентифицирует молекулы, предположительно принимающие участие в воспалении.
5. Система по любому из пп. 1-4, где указанная биологическая функция молекулы в воспалительном процессе выбрана из группы, состоящей из дегрануляции тучных клеток, дифференциации макрофагов, дифференциации миелоидных клеток, дифференциации лимфоцитов, дифференциации иммунных клеток, регуляции гемопоэза, стимуляции врожденного иммунного ответа, путей передачи сигнала рецептора распознавания образов, продуцирования цитокинов, регуляции приобретенного иммунного ответа, опосредованного Т-клетками иммунного ответа, отбора Т-клеток, регуляции пролиферации В-клеток, регуляции секреции иммуноглобулинов, регуляции пролиферации Т-клеток, регуляции пролиферации лимфоцитов, устранения воспаления, регуляции экспрессии генов, модификации белков, развития кровеносных сосудов, иммунного ответа, гемопоэза, развития нейронов и транспорта белков.
6. Система по любому из пп. 1-5, где указанный алгоритм на основе компьютерной программы, включенный в блок обработки, который генерирует карту сети на основе множества наборов данных в базе данных и который позволяет идентифицировать узлы в карте сети, включает правила для приоритизации генов, определения степени узла, определения центральности по посредничеству, идентификации мотива, в частности идентификации петель обратной связи или прямой связи, и/или определения ассоциации с воспалением.
7. Система по п. 1, где наборы данных удаляют из базы данных, если не найдено совпадений между идентификатором молекулы в наборе данных и идентификатором молекулы в транскриптомных данных.
8. Система по п. 1 или 7, где указанным лекарственным средством является многокомпонентное лекарственное средство.
9. Система по п. 8, где указанным многокомпонентным лекарственным средством является траумель.
10. Система по любому из пп. 1-9, где блок визуализации включает алгоритм на основе компьютерной программы, который графически размещает узлы с высокой связью близко друг к другу и/или который идентифицирует для графического отображения только узлы, которые связаны внутримодулярно.
11. Способ моделирования молекулярных взаимодействий, принимающих участие в воспалительном процессе у субъекта, включающий:
(I) обеспечение блока обработки данных, включающего:
(а) базу данных, включающую множество наборов данных, каждый из которых включает:
(i) по меньшей мере один идентификатор молекулы, для которой предполагается участие в патологическом процессе,
(ii) данные по молекулярным взаимодействиям указанной молекулы с одной или более других молекул, и
(iii) по меньшей мере одну характеристику данных, выделенную для набора данных, где каждый набор данных связан по меньшей мере в одном отношении с другим набором данных в базе данных, основанной на молекулярных взаимодействиях;
где наборы данных сгруппированы в секции данных, включающие наборы данных, содержащих идентичные характеристики данных; и
где характеристики данных указывают на биологическую функцию молекулы в воспалительном процессе;
где транскриптомные данные, полученные у субъекта после введения лекарственного средства, также предоставлены в базе данных, и эти транскриптомные данные используют для моделирования наборов данных в базе данных;
и
(б) алгоритм на основе компьютерной программы, включенный в блок обработки данных, который генерирует карту сети, основанную на множестве наборов данных в базе данных и который позволяет идентифицировать узлы в карте сети на основании предварительно определенных параметров; и
(II) генерирование карты сети, основанной на множестве наборов данных в базе данных, и идентификацию узлов в карте сети, основанной на предварительно определенных параметрах в ходе выполнения алгоритма на основе компьютерной программы, реализованного в блоке обработки данных; и
(III) определение молекулярных взаимодействий, принимающих участие в воспалительном процессе, в идентифицированных узлах с использованием блока визуализации, с помощью которого моделируются молекулярные взаимодействия, принимающие участие в воспалительном процессе у субъекта.
12. Способ по п. 11, где указанный воспалительный процесс включает устранение воспаления.
13. Способ по п. 11 или 12, где указанную молекулу идентифицируют из ассоциированных с повреждением образ-распознающих молекул (DAMP) или из патоген-ассоциированных образ-распознающих молекул (РАМР).
14. Способ по п. 13, где указанный DAMP и/или указанный РАМР получают на основе оценки публикаций в общедоступных научных базах данных с использованием алгоритма автоматической оценки, который идентифицирует молекулы, предположительно принимающие участие в воспалении.
15. Способ по любому из пп. 11-14, где указанная биологическая функция молекулы в воспалительном процессе выбрана из группы, состоящей из дегрануляции тучных клеток, дифференциации макрофагов, дифференциации миелоидных клеток, дифференциации лимфоцитов, дифференциации иммунных клеток, регуляции гемопоэза, стимуляции врожденного иммунного ответа, путей передачи сигнала рецептора распознавания образов, продуцирования цитокинов, регуляции приобретенного иммунного ответа, опосредованного Т-клетками иммунного ответа, отбора Т-клеток, регуляции пролиферации В-клеток, регуляции секреции иммуноглобулинов, регуляции пролиферации Т-клеток, регуляции пролиферации лимфоцитов, устранения воспаления, регуляции экспрессии генов, модификации белков, развития кровеносных сосудов, иммунного ответа, гемопоэза, развития нейронов и транспорта белков.
16. Способ по любому из пп. 11-15, где указанное генерирование карты сети на основе множества наборов данных в базе данных и идентификация узлов в карте сети включает приоритизацию генов, определение степени узла, определение центральности по посредничеству, идентификацию мотива, в частности идентификацию петель обратной связи или прямой связи, и/или определение ассоциации с воспалением.
17. Способ по п. 11, где наборы данных удаляют из базы данных, если не найдено совпадений между идентификатором молекулы в наборе данных и идентификатором молекулы в транскриптомных данных.
18. Способ по п. 11 или 17, где указанным лекарственным средством является многокомпонентное лекарственное средство.
19. Способ по п. 18, где указанным многокомпонентным лекарственным средством является траумель.
20. Способ по любому из пп. 11-19, где блок визуализации включает алгоритм на основе компьютерной программы, который графически размещает узлы с высокой связью близко друг к другу и/или который идентифицирует для графического отображения только узлы, которые связаны внутримодулярно.
21. Применение системы по любому из пп. 1-10 для моделирования молекулярных взаимодействий, принимающих участие в воспалительном процессе у субъекта.
22. Применение по п. 21, где моделирование молекулярных взаимодействий, принимающих участие в воспалительном процессе у субъекта, используют для определения действия лекарственного средства.
| JIDAPA SORNSIRI ET AL, "Prediction of biochemical mechanism of anti-inflammation explained from two marine-derived bioactive compounds", AGRICULTURE AND NATURAL RESOURCES,Vol | |||
| Устройство для устранения мешающего действия зажигательной электрической системы двигателей внутреннего сгорания на радиоприем | 1922 |
|
SU52A1 |
| Приспособление для точного наложения листов бумаги при снятии оттисков | 1922 |
|
SU6A1 |
| JINY NAIR ET AL, "Network Analysis of Inflammatory Genes and Their Transcriptional Regulators in Coronary Artery Disease", PLOS ONE,Vol | |||
| Разборный с внутренней печью кипятильник | 1922 |
|
SU9A1 |
| Очаг для массовой варки пищи, выпечки хлеба и кипячения воды | 1921 |
|
SU4A1 |
Авторы
Даты
2023-10-10—Публикация
2020-01-21—Подача