Изобретение относится к области биотехнологии, в частности к определению аллелей гена человека TLR2 по однонуклеотидному полиморфизму rs3804100 в образах биологического материала методом полимеразной цепной реакции (ПЦР) в режиме реального времени и может применяться в исследованиях по изучению влияния однонуклеотидных полиморфизмов (SNP) в генах врожденного иммунитета на возникновение и течение мультифакторных заболеваний, а также в практической деятельности при диагностировании генетических заболеваний, обусловленных таким влиянием.
Толл-подобные рецепторы (TLR) - рецепторы врожденного иммунитета, располагающиеся как на поверхности, так и внутри клетки и распознающие патоген, проникающий в организм. TLR распознают не только экзогенные молекулярные паттерны, связанные с патогеном (PAMPs), но и эндогенные молекулы, связанные с повреждением (DAMPs), которые высвобождаются из поврежденных или умирающих клеток. Активированные TLR с помощью PAMPs или DAMPs инициируют сигнальные каскады, такие как ядерный фактор - каппа В (NFkB), пути митоген-ассоциированной протеинкиназы (MAPK) и интерферона (IFN), и способствуют секреции цитокинов и хемокинов, которые регулируют иммунные и воспалительные реакции, направленные на элиминацию патогенов.
Показана связь SNP в генах TLR с предрасположенностью к возникновению и тяжелому течению мультифакторных заболеваний. Определение аллелей SNP в генах TLR может использоваться для предупреждения тяжелого течения инфекционных заболеваний.
В исследованиях однонуклеотидных полиморфизмов, связанных с туберкулезом легких, наиболее распространены полиморфизмы гена TLR2, что доказывает связь этого рецептора с распознаванием бактерий. Наиболее распространенные SNP, которые связывают с развитием туберкулеза - rs3804099 и rs3804100 (TLR2). В частности, связь rs3804100-C (TLR2) с тяжестью туберкулеза была показана у пациентов с множественной лекарственной устойчивостью. Полиморфизм rs3804100-T (TLR2) связывают с высокой вероятностью возникновения пневмонии на фоне COVID-19.
Из уровня техники известны решения, направленные на выявление специфических нуклеотидных последовательностей полиморфизма rs3804100 в генах TLR2, в частности: комбинации маркеров для оценки риска сепсиса [заявка WO 2008107377, дата подачи заявки 29.02.2008, дата публикации 12.09.2008, дата приоритета 02.03.2007], варианты нуклеиновых кислот в генах толл-подобных рецепторов, ассоциированные с измененным врожденным иммунитетом [заявка WO 2007025989, дата подачи 30.08.2006, дата публикации 08.03.2007, дата приоритета 02.09.2005], способы и средства определения функционального полиморфизма фиколина-2 для прогнозирования исхода трансплантации почки [патент ЕР 2508618, дата подачи заявки 04.04.2011, дата публикации 10.10.2012]. Упомянутые решения реализуются в формате мультиплекс, определяют полиморфизм rs3804100 гена TLR2 среди прочих, и их применение для целей генотипирования указанного полиморфизма является затратным и малоинформативным.
Известны наборы реагентов для определения аллелей SNP методом ПЦР в режиме реального времени. Наборы реагентов включают в себя олигонуклеотиды для детекции нескольких специфичных аллелей в исследуемых образцах и отличаются удобством для пользователя. Для выбранных мишеней детекции не всегда подтверждена связь с заболеванием и поэтому такие наборы не всегда могут использоваться в клинической практике, но используются в научно-исследовательских целях. Из уровня техники также известен набор реагентов «ThermoFisher Scientific С_30687121_20» для определения аллелей rs5743565 гена TLR1 [https://www.thermofisher.com/order/genome-database/details/genotyping/C_30687121_20?CID=&ICID=&subtype=#more-information-section]. Однако данное решение не предназначено для определения аллелей rs3804100 гена TLR2.
Также генотипирование локусов rs3804100 выполняли с применением реагентов для выделения ДНК («РИБО-преп»), амплификации и пробоподготовки («ПИРО-преп») производства Центрального НИИ эпидемиологии Роспотребнадзора («АмплиСенс», Россия) методом пиросеквенирования с использованием реагентов PyroMark Q96 и системы генетического анализа PyroMark Q24 («Qiagen», Германия) [М.А. Карнаушкина, А.С.Гурьев, К.О. Миронов, Е.А. Дунаева, В.И. Корчагин, О.Ю. Бобкова, И.С. Васильева, Д.В. Кассина, М.М. Литвинова. Ассоциации полиморфизмов генов толл-подобных рецепторов и активности нетоза как прогностические критерии тяжести течения пневмонии. http://www.stm-journal.ru/ru/numbers/2021/3/1723/html]. Согласно описанному способу были определены rs3804100-C и rs3804100-T гена TLR2 методом пиросеквенирования, который делает его более затратным и трудоемким.
Таким образом, существует потребность в расширении точного диагностического инструментария, позволяющего в короткие сроки и с наименьшими затратами генотипировать аллели гена TLR2 по полиморфизму rs3804100 для своевременного и адекватного назначения диагностических, терапевтических и профилактических мероприятий, а также для дальнейшего изучения врожденного иммунитета и механизма возникновения и течения мультифакторных заболеваний.
Технический результат заявляемого изобретения направлен на генотипирование аллелей С и Т гена TLR2 по полиморфизму rs3804100, а именно определение rs3804100-C и rs3804100-T гена TLR2, в образцах биологического материала с использованием широко распространенных методик и доступного оборудования.
Заявленный технический результат достигается за счет проведения полимеразной цепной реакции с гибридизационно-флуоресцентной детекцией продуктов амплификации в режиме реального времени с применением реакционной смеси, содержащей набор уникальных синтезированных нуклеотидных праймеров и конформационно-блокированных зондов, а генотипирование осуществляют посредством оценки кривых накопления флуоресцентных сигналов по соответствующим каналам для флуорофоров, а именно: для rs3804100-C по каналу FAM, для rs53804100-T по каналу R6G.
Упомянутый набор синтезированных нуклеотидных праймеров и зондов позволяет эффективно детектировать полиморфный нуклеотид rs3804100 в последовательности ДНК и имеет следующий олигонуклеотидный состав:
прямой праймер 4100-F - SEQ ID NO: 1,
обратный праймер 4100-R - SEQ ID NO: 2,
флуоресцентный зонд 4100-С - SEQ ID NO: 3,
флуоресцентный зонд 4100-Т - SEQ ID NO: 4.
Праймеры представляют собой последовательности олигонуклеотидов для амплификации фрагмента, включающей полиморфизм rs3804100, флуоресцентные зонды являются олигонуклеотидами, содержащими флуорофор, гаситель флуоресценции, позволяющими детектировать аллели в амплифицированный фрагмент.
Заявляемый набор олигонуклеотидных праймеров и зондов разработан на основе проведенного анализа последовательностей: гена TLR2 toll like receptor 2 (Homo sapiens (human)), Gene ID: 7097 NC_000004.12 GRCh38.p14 (GCF_000001405.40), представленных в базе данных National Center for Biotechnology Information (NCBI) [NCBI https://www.ncbi.nlm.nih.gov/gene/7097], и не имеющих гомологии с другими последовательностями ДНК. В результате выбран фрагмент для детекции участка, содержащего rs3804100 гена TLR2 к которому подобраны прямой и обратный праймеры для амплификации фрагмента длиной 95 пар оснований, а также флуоресцентные зонды для детекции аллелей С и Т выбранного SNP: прямой праймер 4100-F - SEQ ID NO: 1; обратный праймер 4100-R - SEQ ID NO: 2; флуоресцентный зонд 4100-С - SEQ ID NO: 3; флуоресцентный зонд 4100-Т - SEQ ID NO: 4.
При этом зонды содержат конформационно-блокированные нуклеотиды, которые позволяют повысить стабильность дуплекса с ДНК-мишенью. Флуоресцентный зонд 4100-С содержит 5 блокаторов, которые расположены в положении 2, 4, 8, 12, 14. Флуоресцентный зонд 4100-Т содержит 6 блокаторов, которые расположены в положении 2, 4, 8, 10, 12, 14.
Для подбора мишеней - мест посадки олигонуклеотидов используют фрагменты референсного генома, взятые из базы данных NCBI dbSNP [NCBI dbSNP https://www.ncbi.nlm.nih.gov/snp/обращено 12.12.2022]. Для поиска гомологичных областей применяют современные алгоритмы in silico анализа нуклеотидных последовательностей и программы, находящиеся в открытом доступе: Primer BLAST, Unipro UGENE. Состав набора олигонуклеотидных праймеров и зондов приведен в Таблице 1.
Анализ результатов продемонстрировал, что прямой праймер 4100-F (SEQ ID NO: 1) и обратный праймер 4100-R (SEQ ID NO: 2) амплифицируют участок гена человека TLR2, содержащий полиморфизм rs3804100 со 100% специфичностью. Также специфичность прямого и обратного праймеров подтверждена с помощью прямого метода пиросеквенирования нуклеотидных последовательностей.
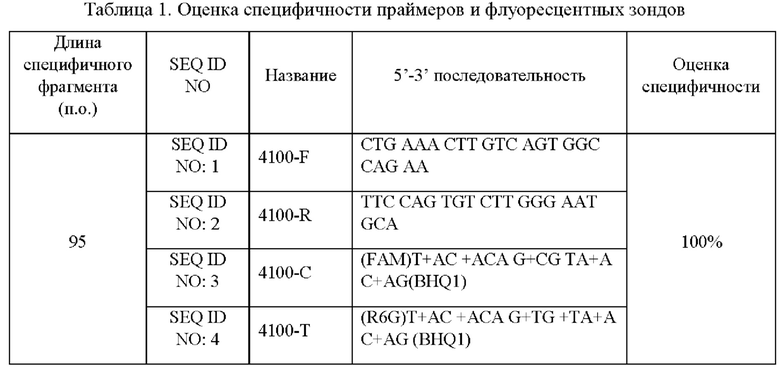
где FAM - флуорофор для SEQ ID NO: 3, R6G - флуорофор для SEQ ID NO: 4, BHQ1 - гаситель флуоресценции, соответствующие красителю каналов флуороворов FAM и R6G, знак «+» перед конформационно-блокированным нуклеотидом.
Заявляемое изобретение является результатом научно-исследовательской работы лаборатории молекулярных методов изучения генетических полиморфизмов «Изучение генетической предрасположенности к мультифакторным заболеваниям», проведенной ФБУН ЦНИИ Эпидемиологии Роспотребнадзора (Москва, Россия).
В качестве биологического материала предпочтительно использовать венозную кровь.
ПЦР - исследование проводят в соответствии с МУ 1.3.2569-09 «Организация работ лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для экстракции ДНК может быть использован комплект реагентов «РИБО-преп» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) или любой аналогичный набор/комплект реагентов для выделения ДНК, в соответствии с инструкцией производителя. Оптимальная концентрация ДНК - 103-105 копий в 10 мкл. ПЦР в режиме реального времени проводится с применением заявляемого набора олигонуклеотидных праймеров и зондов для детекции аллелей полиморфизма rs3804100 гена человека TLR2, представленных в Таблице 1.
ПЦР проводят при следующих условиях:
Общий объем реакционной смеси - 25 мкл.
Компоненты ПЦР смешиваются следующим образом:
(a) 10 мкл смеси, содержащей:
- олигонуклеотидные праймеры SEQ ID NO NO: 1, 2 - по 0,07 мМ;
- флуоресцентные зонды SEQ ID NO NO: 3, 4 - по 0,02 мМ
- dNTPs - 0,44 мМ.
(b) реактив, содержащий рекомбинантный фермент Taq ДНК-полимеразу, например, 0,5 мкл «Полимераза TaqF» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Москва, Россия) или любого аналогичного коммерческого набора в соответствии с инструкцией производителя.
(c) ПЦР-буфер, содержащий MgCl2, например, 5,0 мкл ПЦР-буфера «ОТ-ПЦР-смесь-2 FEP/FRT» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Москва, Россия) или любого аналогичного коммерческого набора в соответствии с инструкцией производителя.
(d) экстрагированная из цельной крови человека ДНК - 10 мкл. Амплификацию проводят на амплификаторе с возможностью детекции флуоресцентного сигнала как минимум по 2 каналам флуоресценции, например, «Rotor-Gene Q» («Qiagen», Германия) в соответствии с инструкцией производителя.
Амплификацию проводят по следующей программе: 1 цикл 95°С в течение 15 минут, 45 циклов при температуре 95°С - 10 секунд / 60°С - 20 секунд. Детекция флуоресценции проводится на этапе 60°С по каналу для флуорофора FAM для rs3804100-С, по каналу для флуорофора R6G - для rs3804100-T.
Анализ данных проводится на основе детекции амплификатором уровня флуоресцентного сигнала. Результаты интерпретируются на основании наличия (или отсутствия) пересечения кривой флуоресценции S-образной (сигмообразной) формы с установленной на соответствующем уровне пороговой линией. Это определяет наличие или отсутствие для анализируемого образца значения порогового цикла. В процессе амплификации ДНК зонды конкурируют за связывание с матрицей ДНК, содержащем два праймера SEQ ID NO: 1 и SEQ ID NO: 2. При этом преимущество получает зонд, соответствующий представленному в образце аллели. Если присутствуют два аллеля полиморфизма, гибридизуются оба зонда.
Для каждого канала задают пороговую линию, соответствующую величине 10% от максимального сигнала флуоресценции образца среди образцов с определенным аллелем. Образцы, для которых кривые флуоресценции пересекают пороговую линию по каналу для детекции флуорофора FAM и при этом кинетика накопления флуоресцентного сигнала является экспоненциальной, содержат аллель С полиморфизма rs3804100 гена TLR2. Образцы, для которых кривые флуоресценции пересекают пороговую линию по каналу для детекции флуорофора R6G и при этом кинетика накопления флуоресцентного сигнала является экспоненциальной, содержат аллель Т полиморфизма rs3804100 гена TLR2.
Образцы, для которых кривые флуоресценции пересекли пороговые линии по каналам для детекции флуорофоров FAM и R6G и при этом кинетика накопления флуоресцентного сигнала является экспоненциальной, содержат rs3804100-C и rs3804100-Т.
Реализация заявляемого изобретения поясняется следующими примерами: Пример 1. Получение набора олигонуклеотидных праймеров и зондов для генотипирования аллелей гена TLR2 по полиморфизму rs3804100
Для подбора целевых последовательностей - мест посадки олигонуклеотидов, использовали фрагменты гена TLR2 toll like receptor 2 (Homo sapiens (human)), Gene ID: 7097 NC_000004.12 GRCh38.p14 (GCF_000001405.40), представленных в базе данных National Center for Biotechnology Information (NCBI) [NCBI https://www.ncbi.nlm.nih.gov/gene/7097], и не имеющих гомологии с другими последовательностями ДНК. Для поиска гомологичных областей использовали современные алгоритмы in silico анализа нуклеотидных последовательностей и программы Primer BLAST, Unipro UGENE, MEGA.
В результате проведенного анализа выбран участок гомологичной последовательности, к которому подобрали праймеры и зонды для амплификации фрагмента длиной 95 пар оснований, а также флуоресцентные зонды для детекции аллелей С и Т выбранного SNP: прямой праймер 41001-F - SEQ ID NO: 1; обратный праймер 4100-R - SEQ ID NO: 2; флуоресцентный зонд 4100-С - SEQ ID NO: 3; флуоресцентный зонд 4100-Т - SEQ ID NO: 4. При этом каждый зонд содержат конформационно заблокированные нуклеотиды, а именно: флуоресцентный зонд 4100-С содержит 5 блокаторов, которые расположены в положении 2, 4, 8, 12, 14, а флуоресцентный зонд 4100-Т - 6 блокаторов, которые расположены в положении 2, 4, 8, 10, 12, 14.
Анализ упомянутых последовательностей с использованием ресурса Primer BLAST [https://www.ncbi.nlm.nih.gov/tools/primer-blast/] показал, что прямой 4100-F и обратный 4100-R праймеры амплифицируют участок, содержащий полиморфизм rs3804100 в гене TLR2 со 100% специфичностью.
Пример 2. Детекция аллелей С и Т гена TLR2 по полиморфизму rs3804100 методом ПЦР в режиме реального времени.
ПЦР в режиме реального времени проводили при следующих условиях.
Общий объем реакционной смеси - 25 мкл.
Компоненты ПЦР смешиваются следующим образом:
(a) 10 мкл смеси, содержащей:
- олигонуклеотидные праймеры: SEQ ID NO:1, SEQ ID NO: 2 по 0,07 мМ;
- флуоресцентные зонды SEQ ID NO: 3, SEQ ID NO: 4 - по 0,02 мМ;
- dNTPs - 0,44 мМ.
(b) 0,5 мкл реактива «Полимераза TaqF» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия), содержащего рекомбинантный фермент Taq ДНК-полимеразу.
(c) 5,0 мкл ПЦР-буфера «ОТ-ПЦР-смесь-2 FEP/FRT» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия), содержащего MgCl2;
(d) экстрагированная из цельной венозной крови человека ДНК - 10 мкл.
ПЦР в режиме реального времени проводили с флуоресцентной детекцией результата на приборе с 5 каналами детекции - «Rotor-Gene Q» («Qiagen», Германия).
Подготовленный описанным способом материал, содержащий уникальные олигонуклеотидные последовательности SEQ ID NO: 1-4, использовали для определения аллелей rs3804100 гена TLR2 в образцах биологического материала.
Пример 3. Генотипирование аллелей С и Т гена TLR2 по полиморфизму rs3804100 в образцах биологического материала.
Для определения аллелей rs3804100 гена человека TLR2 в образцах биологического материала выбрано 71 проба, для исследования использовали биологический материал - венозная кровь. Выделение ДНК проводили в соответствии с МУ 1.3.2569-09 «Организация работ лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для экстракции ДНК использовали набор реагентов «РИБО-преп» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) в соответствии с инструкцией производителя.
ПЦР-РРВ проводили в условиях, описанных в Примере 2, с использованием уникальных синтезированных олигонуклеотидных праймеров и зондов SEQ ID NO: 1-4.
Амплификацию проводили на приборе «Rotor-Gene Q» («Qiagen», Германия) по следующей программе: 1 цикл 95°С в течение 15 минут, 45 циклов при температуре 95°C - 10 секунд / 60°С - 20 секунд. Детекция флуоресцентного сигнала проводилась на этапе 60°С по каналу для флуорофора FAM для алелля С, и по каналу для флуорофора R6G для алелля Т.
При анализе результатов для каждого канала задали пороговую линию, соответствующую величине 10% от максимального сигнала флуоресценции образца среди образцов с определенным аллелем.
Для 13/71 образцов кривая флуоресценции пересекла пороговую линию по каналу для флуорофора FAM, для 58/71 образцов кривая флуоресценции пересекла пороговую линию по каналу для флуорофора R6G, при этом кинетика накопления флуоресцентных сигналов была экспоненциальной.
По каналу для флуорофора FAM определили наличие аллеля rs3804100-C, по каналу для флуорофора R6G определили наличие аллеля rs3804100-T полиморфизма.
Результаты исследования были подтверждены с помощью метода пиросеквенирования нуклеотидных последовательностей, которое выполнялось посредством системы генетического анализа PyroMark Q24 («Qiagen», Германия) в соответствии с инструкцией производителя.
Пример 4. Генотипирование аллелей С и Т гена TLR2 по полиморфизму rs3804100 в образцах биологического материала.
Для определения аллелей rs3804100 гена человека TLR2 в образцах биологического материала выбрано 11 проб, для исследования использовали биологический материал - цельная кровь. Выделение ДНК проводили в соответствии с МУ 1.3.2569-09 «Организация работ лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для экстракции ДНК использовали набор реагентов «РИБО-преп» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) в соответствии с инструкцией производителя.
ПЦР-РРВ проводили в условиях, описанных в Примере 2, с использованием уникальных синтезированных олигонуклеотидных праймеров и зондов SEQ ID NO: 1-4.
Амплификацию проводили на приборе «Rotor-Gene Q» («Qiagen», Германия) по следующей программе: 1 цикл 95°С в течение 15 минут, 45 циклов при температуре 95°С - 10 секунд / 60°С - 20 секунд. Детекция флуоресцентного сигнала проводилась на этапе 60°С по каналу для флуорофора FAM - для rs3804100-C, и по каналу для флуорофора R6G - для rs3804100-T.
При анализе результатов для каждого канала задали пороговую линию, соответствующую величине 10% от максимального сигнала флуоресценции образца среди образцов с определенным аллелем.
Для 11/11 образцов кривая флуоресценции пересекла пороговую линию по каналу для флуорофора FAM, что позволило определить наличие аллеля С полиморфизма rs3804100 гена TLR2 в 11 образцах из выборки.
Результаты исследования были подтверждены с помощью метода пиросеквенирования нуклеотидных последовательностей, которое выполнялось посредством системы генетического анализа PyroMark Q24 («Qiagen», Германия) в соответствии с инструкцией производителя.
Пример 5. Генотипирование аллелей С и Т гена TLR2 по полиморфизму rs3804100 в образцах биологического материала.
Для определения аллелей rs3804100 гена человека TLR2 в образцах биологического материала выбрано 16 проб, для исследования использовали биологический материал - венозная кровь. Выделение ДНК проводили в соответствии с МУ 1.3.2569-09 «Организация работ лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для экстракции ДНК использовали набор реагентов «РИБО-преп» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) в соответствии с инструкцией производителя.
ПЦР-РРВ проводили в условиях, описанных в Примере 2, с использованием уникальных синтезированных олигонуклеотидных праймеров и зондов SEQ ID NO: 1-4.
Амплификацию проводили на приборе «Rotor-Gene Q» («Qiagen», Германия) по следующей программе: 1 цикл 95°С в течение 15 минут, 45 циклов при температуре 95°С - 10 секунд / 60°С - 20 секунд. Детекция флуоресцентного сигнала проводилась на этапе 60°С по каналу для флуорофора FAM для rs3804100-C, и по каналу для флуорофора R6G для rs3804100-T.
При анализе результатов для каждого канала задали пороговую линию, соответствующую величине 10% от максимального сигнала флуоресценции образца среди образцов с определенным аллелем.
Для 16/16 образцов кривая флуоресценции пересекла пороговую линию по каналу для флуорофора R6G, что позволило определить наличие аллеля Т полиморфизма rs3804100 гена TLR2 в 16 образцах из выборки.
Результаты исследования были подтверждены с помощью метода пиросеквенирования нуклеотидных последовательностей, которое выполнялось посредством системы генетического анализа PyroMark Q24 («Qiagen», Германия) в соответствии с инструкцией производителя.
Пример 6. Генотипирование аллелей С и Т гена TLR2 по полиморфизму rs3804100 в образцах биологического материала.
Для определения аллелей rs3804100 гена человека TLR2 в образцах биологического материала выбрано 52 пробы, для исследования использовали биологический материал - цельная кровь. Выделение ДНК проводили в соответствии с МУ 1.3.2569-09 «Организация работ лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для экстракции ДНК использовали набор реагентов «РИБО-преп» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) в соответствии с инструкцией производителя.
ПЦР в режиме реального времени проводили в условиях, описанных в Примере 2, с использованием уникальных синтезированных олигонуклеотидных праймеров и зондов SEQ ID NO: 1-4.
Амплификацию проводили на приборе «Rotor-Gene Q» («Qiagen», Германия) по следующей программе: 1 цикл 95°С в течение 15 минут, 45 циклов при температуре 95°С - 10 секунд / 60°С - 20 секунд. Детекция флуоресцентного сигнала проводилась на этапе 60°С по каналу для флуорофора FAM - для алелля С, и по каналу для флуорофора R6G - для алелля Т.
При анализе результатов для каждого канала задали пороговую линию, соответствующую величине 10% от максимального сигнала флуоресценции образца среди образцов с определенным аллелем.
Для 11/52 образцов кривая флуоресценции пересекла пороговую линию по каналу для флуорофора FAM, для 29/52 образцов кривые флуоресценции пересекли пороговую линию по каналу для флуорофора R6G, для 12/37 образцов кривая флуоресценции пересекла пороговые линии по каналам для флуорофора FAM и R6G, при этом кинетика накопления флуоресцентных сигналов была экспоненциальной.
По каналу для флуорофора FAM определили наличие ДНК rs3804100-C, по каналу для флуорофора R6G определили наличие ДНК rs3804100-T, наличие сигналов по двум каналам для флуорофоров FAM и R6G одновременно подтвердили присутствие ДНК двух аллелей - rs3804100-C и rs3804100-T.
Результаты исследования были подтверждены с помощью метода пиросеквенирования нуклеотидных последовательностей, которое выполнялось посредством системы генетического анализа PyroMark Q24 («Qiagen», Германия) в соответствии с инструкцией производителя.
Таким образом, заявляемое изобретение позволяет детектировать аллели rs3804100-C и rs3804100-T гена TLR2 в образцах биологического материала. Набор уникальных синтезированных олигонуклеотидных праймеров и зондов SEQ ID NO: 1-4 не дает перекрестных реакций с другими последовательностями генома, амплифицирует последовательность, включающую rs3804100, со 100% специфичностью и позволяют однозначная интерпретировать полученные результаты.
--->
<ST26SequenceListing dtdVersion="V1_2" fileName="rs3804100"
softwareName="WIPO Sequence" softwareVersion="1.0.0"
productionDate="2023-04-12">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>no</ApplicationNumberText>
<FilingDate>2023-04-11</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>01</ApplicantFileReference>
<ApplicantName languageCode="ru">ФБУН ЦНИИ Эпидемиологии
Роспотребнадзора</ApplicantName>
<ApplicantNameLatin>FBUN CRIE</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ генотипирования гена TLR2 по
полиморфизму rs3804100 и набор олигонуклеотидных праймеров и зондов
для его реализации</InventionTitle>
<SequenceTotalQuantity>4</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>rs3804100</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctgaaacttgtcagtggccagaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>rs3804100</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttccagtgtcttgggaatgca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>rs3804100</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tacacagcgtaacag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>rs3804100</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tacacagtgtaacag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ генотипирования гена TLR2 по полиморфизму rs5743708 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805861C1 |
| Способ генотипирования гена TLR4 по полиморфизму rs4986790 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805862C1 |
| Способ генотипирования гена TLR6 по полиморфизму rs5743810 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805863C1 |
| Способ генотипирования гена TLR8 по полиморфизму rs3764880 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805864C1 |
| Способ генотипирования гена TLR1 по полиморфизму rs5743551 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805859C1 |
| Набор олигонуклеотидных праймеров и зондов для определения аллей полиморфизма rs55986091 и способ его применения | 2022 |
|
RU2804110C1 |
| Набор олигонуклеотидных праймеров и зондов для видовой дифференциации вируса герпеса человека 6А и вируса герпеса человека 6В и способ его применения | 2022 |
|
RU2806427C1 |
| Мультиплексная ПЦР-смесь для определения серотипов 12FAB, 15BC, 22FA, 8 Streptococcus pneumoniae и способ ее применения | 2021 |
|
RU2787181C1 |
| Способ количественного определения ДНК вируса герпеса человека 6А (ВГЧ-6А) и ДНК вируса герпеса человека 6В (ВГЧ-6В) и набор олигонуклеотидных праймеров и зонда для его реализации | 2022 |
|
RU2800731C1 |
| Способ количественного определения ДНК вируса герпеса человека 6А (ВГЧ-6А) и набор олигонуклеотидных праймеров и зонда для его реализации | 2022 |
|
RU2802931C1 |
Изобретение относится к биотехнологии. Предложен способ генотипирования аллелей С и Т гена TLR2 по полиморфизму rs3804100, включающий: экстракцию ДНК из биологического материала, проведение ПЦР в режиме реального времени с применением реакционной смеси, содержащей набор нуклеотидных праймеров и зондов SEQ ID NO: 1-4, амплификацию, анализ и интерпретацию результатов, где наличие аллеля С гена TLR2 по полиморфизму rs3804100 определяется посредством оценки кривых накопления флуоресцентных сигналов по каналу для флуорофора FAM, а наличие в образце биологического материала аллели T гена TLR2 по полиморфизму rs3804100 определяется посредством оценки кривых накопления флуоресцентных сигналов по каналу для флуорофора R6G. Предложен набор праймеров для осуществления заявленного способа. Изобретение позволяет детектировать аллели rs3804100-C и rs3804100-T гена TLR2 в образах биологического материала методом ПЦР в режиме реального времени. 2 н. и 3 з. п. ф-лы, 1 табл., 6 пр.
1. Способ генотипирования аллелей С и Т гена TLR2 по полиморфизму rs3804100, включающий: экстракцию ДНК из биологического материала, проведение ПЦР в режиме реального времени с применением реакционной смеси, содержащей набор нуклеотидных праймеров и зондов SEQ ID NO: 1-4, амплификацию, анализ и интерпретацию результатов, где наличие аллеля С гена TLR2 по полиморфизму rs3804100 определяется посредством оценки кривых накопления флуоресцентных сигналов по каналу для флуорофора FAM, а наличие в образце биологического материала аллели T гена TLR2 по полиморфизму rs3804100 определяется посредством оценки кривых накопления флуоресцентных сигналов по каналу для флуорофора R6G.
2. Способ генотипирования по п. 1, где наличие в образце биологического материала аллеля С гена TLR2 по полиморфизму rs3804100 и аллеля T гена TLR2 по полиморфизму rs3804100 определяется посредством оценки кривых накопления флуоресцентных сигналов по каналам FAM и R6G одновременно.
3. Способ генотипирования по пп. 1 и 2, где для анализа результатов задают пороговую линию, соответствующую величине 10% от максимального сигнала флуоресценции образца среди образцов с определенным аллелем.
4. Набор олигонуклеотидных праймеров и зондов для применения в способе генотипирования по пп. 1 и 2, имеющий следующий нуклеотидный состав:
прямой праймер 4100-F - SEQ ID NO: 1;
обратный праймер 41001-R - SEQ ID NO: 2;
флуоресцентный зонд 4110-С - SEQ ID NO: 3;
флуоресцентный зонд 4100-Т - SEQ ID NO: 4,
при этом флуоресцентный зонд 4100-С содержит конформационно-блокированные нуклеотиды, расположенные в положении 2, 4, 8, 12, 14, а флуоресцентный зонд 4100-T содержит конформационно-блокированные нуклеотиды, расположенные в положении 2, 4, 8, 10, 12, 14.
5. Набор олигонуклеотидных праймеров и зондов по п. 4, где для создания прямого и обратного праймеров, флуоресцентных зондов используют фрагмент гена TLR2 Homo sapiens.
| WO 2008107377 А2, 12.09.2008 | |||
| WO 2007025989 А2, 08.03.2007 | |||
| М.А | |||
| Карнаушкина и др | |||
| Ассоциации полиморфизмов генов толл-подобных рецепторов и активности нетоза как прогностические критерии тяжести течения пневмонии, том 13, номер 3 (2021), весь документ, http://www.stm-journal.ru/ru/numbers/2021/3/1723/html | |||
| Барбараш О.Л | |||
| и др | |||
| РОЛЬ ПОЛИМОРФИЗМА |
Авторы
Даты
2023-10-24—Публикация
2023-04-13—Подача