Изобретение относится к области биотехнологии, в частности к определению полиморфизма rs55986091 между генами HLA-DQA1 и HLA-DQB1 с использованием полимеразной цепной реакции в режиме реального времени (ПЦР-РРВ) и может использоваться для определения к предрасположенности к раку шейки матки (РШМ) и другим онкологическим заболеваниям, ассоциированным с вирусом папилломы человека.
Рак шейки матки является четвертым по встречаемости и смертности раком в мире среди женщин: в 2020 году зарегистрировано 604 тысяч новых случаев и более 340 тысяч смертельных исходов от данной нозологии, что составляет 7,7% всех смертей, связанных с злокачественными новообразованиями. Число случаев РШМ в России растет: за 10 лет (с 2009 по 2019 год) увеличилось практически на 22% (с 14 до 17 тысяч), что демонстрирует социальную значимость данного заболевания. Особого внимания заслуживает факт, что на возрастную группу женщин 30-44 года, то есть социально активного и репродуктивного возраста, приходится 32,4% случаев РШМ. Доказанным канцерогенным фактором РШМ является инфицирование вирусом папилломы человека (ВПЧ) высокого канцерогенного риска (ВПЧ ВКР). При этом у ВПЧ-положительных женщин частота РШМ составляет всего 0,015%, что позволяет предполагать о наличие генетической предрасположенности. Определение однонуклеотидных полиморфизмов, ассоциированных с заболеваниями, позволяет охарактеризовать возможную наследственную предрасположенность к развитию патологических состояний в досимптоматический период для своевременного назначения диагностических или профилактических мероприятий.
Ранее Bowden и соавторы в GWAS - исследовании по полногеномному скринингу ассоциаций - показали устойчивую ассоциацию rs55986091 с РШМ, для определения предрасположенности к РШМ использован метод детекции генотипа с помощью биочипов [Bowden SJ, Bodinier В, Kalliala I, Zuber V, Vuckovic D, Doulgeraki T, Whitaker MD, Wielscher M, Cartwright R, Tsilidis KK, Bennett P, Jarvelin MR, Flanagan JM, Chadeau-Hyam M, Kyrgiou M; FinnGen consortium. Genetic variation in cervical preinvasive and invasive disease: a genome-wide association study. Lancet Oncol. 2021; 22(4):548-557.]. К недостаткам метода можно отнести дорогостоящее производство реагентов и низкую доступность/представленность соответствующего оборудования в отечественных лабораториях, высокую сложность и длительность предварительной подготовки образцов к проведению исследования.
Оптимальным и быстрым способом получения результатов с низкой сложностью предварительной подготовки образцов для определения аллелей rs55986091 является ПЦР-РРВ с использованием флуоресцентно меченых зондов.
Из уровня техники известна методика для определения генетических факторов, ассоциированных с предрасположенностью к РШМ где в формате ПЦР-РРВ определяют аллелеи и генотипы rs138446575 и rs2910164. Определены следующие частоты генотипы в выборке 138 человек rs2910164: GG - 83, GC - 49 и СС - 6; rs138446575: СС - 134 и СТ - 4. Сравнение частот аллелей в данной выборке не выявило статистических значимых отличий с выборкой в базе данных Ensembl для европеоидов (р>0,2) и соответствовало распределению Харди-Вайнберга (р>0,1) [Винокуров М.А., Миронов К.О. Разработка методики определения генетических полиморфизмов, ассоциированных с раком шейки матки. Молекулярная диагностика и биобезопасность-2022. Конгресс с международным участием (Москва, 27-28 апреля 2022 года). Сборник материалов; Москва, ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, 2022, стр. 184-185]. Данные методики позволяют детектировать другие полиморфизмы, ассоциированные с РШМ, методом ПЦР-РРВ. При этом схожих методик для определения rs55986091 методом ПЦР-РРВ не выявлено.
Таким образом, существует потребность в расширении точного диагностического решения, позволяющего в короткие сроки и с наименьшими затратами определять полиморфизм rs55986091 у женщин для своевременного назначения диагностических или профилактических мероприятий.
Детекция полиморфизма rs55986091 заключается в определении замены G>A, при этом аллель G ассоциирован с предрасположенностью к РШМ. Соответственно аллель А является протективным. Рассчитанные р-значения для показателя отношения шансов, полученные на большой выборке (5 тысяч больных РШМ), демонстрируют актуальность заявляемого решения.
Технический результат заявляемого изобретения направлен на выявление аллеля G или А в образцах биологического материала посредством набора олигонуклеотидных праймеров и флоуресцентных зондов с LNA модификацией, которые позволяют эффективно детектировать полиморфный нуклеотид в последовательность ДНК с использованием широко распространенных методик и доступного оборудования.
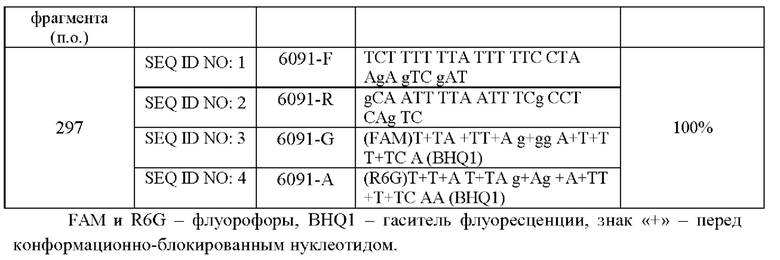
Технический результат достигается за счет применения в ПЦР-РРВ набора синтезированных праймеров и зондов для определения полиморфизма rs55986091, имеющих следующий олигонуклеотидный состав:
прямой праймер 6091-F SEQ ID NO: 1;
обратный праймер 6091-R - SEQ ID NO: 2;
флоуресцентный зонд 6091-G - SEQ ID NO: 3.
флоуресцентный зонд 6091-А - SEQ ID NO: 4.
Праймеры представляют собой последовательности олигонуклеотидов для амплификации фрагмента ДНК, включающей полиморфизм rs55986091, флуоресцентные зонды являются олигонуклеотидами, содержащим флуорофор, гаситель флуоресценции, позволяющим детектировать аллели в амплифицированный фрагмент.
Заявляемый набор олигонуклеотидных праймеров и зондов разработан на основе данных S.J. Bowden и соавторов, которые выявили локус между генами HLA-DQA1 и HLA-DQB1, для разработки диагностической системы [Bowden SJ, Bodinier В, Kalliala I, Zuber V, Vuckovic D, Doulgeraki T, Whitaker MD, Wielscher M, Cartwright R, Tsilidis KK, Bennett P, Jarvelin MR, Flanagan JM, Chadeau-Hyam M, Kyrgiou M; FinnGen consortium. Genetic variation in cervical preinvasive and invasive disease: a genome-wide association study. Lancet Oncol. 2021; 22(4):548-557]. С этой целью были проанализирован фрагмент последовательности генома, включающий rs55986091, и не имеющий гомологии с другими последовательностями ДНК. В результате проведенного анализа выбран участок гомологичной последовательности, к которому подобрали праймеры и зонды для амплификации фрагмента размером 297 пар оснований: прямой 6091-F - SEQ ID NO: 1; обратный 6091-R - SEQ ID NO: 2; флоуресцентный зонд 6091-G - SEQ ID NO: 3 и флоуресцентный зонд 6091-А - SEQ ID NO: 4.
Для подбора мишеней - мест посадки олигонуклеотидов используют фрагменты референсного генома, взятые из базы данных NCBI dbSNP [NCBI dbSNP https://www.ncbi.nlm.nih.gov/snp/ обращено 10.09.2021]. Для поиска гомологичных областей применяют современные алгоритмы in silico анализа нуклеотидных последовательностей и программы, находящиеся в открытом доступе: Primer BLAST, Unipro UGENE. Состав набора олигонуклеотидных последовательностей и зондов приведен в Таблице 1.
Анализ заявляемых последовательностей с использованием ресурса Primer BLAST продемонстрировал, что прямой 6091-F (SEQ ID NO: 1) и обратный 6091-R (SEQ ID NO: 2) праймеры амплифицируют участок ДНК, включающий rs55986091, со 100% специфичностью. Также специфичность прямого и обратного праймеров подтверждена с помощью секвенирования нуклеотидных последовательностей.

Для детекции аллеля G в образцах используют канал для флуорофора FAM, для аллеля А канал для флуорофора R6G.
Заявляемое изобретение является результатом работы в рамках исследования генетической предрасположенности к РШМ, проделанной в ФБУН ЦНИИ Эпидемиологии Роспотребнадзора (Москва, Россия).
ПЦР-исследование проводят в соответствии с МУ 1.3.2569-09 «Организация работ лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для экстракции ДНК может быть использован комплект реагентов «РИБО-преп» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) или любой аналогичный набор/комплект реагентов для выделения ДНК. Оптимальная концентрация ДНК - 10-30 нг в 10 мкл (соответствует концентрации выделенной ДНК 3×105 - 9×105 копий/мл) ПЦР-РРВ проводится с применением заявляемого набора олигонуклеотидных праймеров и зондов для детекции rs55986091, представленных в Таблице 1.
ПЦР-РРВ проводят при следующих условиях:
Общий объем реакционной смеси - 25 мкл.
Компоненты ПЦР смешиваются следующим образом:
(a) 10 мкл смеси, содержащей:
- олигонуклеотидные праймеры SEQ ID NO: 1, 2 - по 0,6 мкМ;
- флоуресцентный зонд SEQ ID NO: 3-0,12 мкМ;
- флоуресцентный зонд SEQ ID NO: 4-0,12 мкМ;
- dNTPs - 0,44 мМ.
(b) реактив, содержащий рекомбинантный фермент Taq ДНК-полимеразы, например, 0,5 мкл реактива «Полимераза TaqF» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) или любого аналогичного коммерческого набора в соответствии с инструкцией производителя.
(c) ПЦР-буфер, содержащий MgCl2, например, 5 мкл ПЦР-буфера «ОТ-ПЦР-смесь-2 FEP/FRT» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) или любого аналогичного коммерческого набора в соответствии с инструкцией производителя.
(d) выделенная ДНК - 10 мкл.
ПЦР-РРВ проводят на программируемом амплификаторе с системой детекции флуоресцентного сигнала в режиме реального времени, имеющем два или более канала флуоресцентной детекции (например, «Rotor-Gene Q» («Qiagen», Германия), «CFX96» («Bio-Rad Laboratories, Inc.», США) и другие.
Амплификацию проводят по следующей программе: 1 цикл 95°С в течение 15 минут, 45 циклов при температуре 95°С - 10 секунд / 60°С - 20 секунд / 72°С - 20 секунд. Детекция флуоресценции проводится на этапе 60°С по каналу для флуорофора FAM - для аллеля G, по каналу для флуорофора R6G - для аллеля А.
Для анализа результатов составляется график количественного анализа, где по оси абсцисс откладывается количество циклов, а по оси ординат уровень флуоресценции. Для каждого образца высчитывается кривая амплификации путем вычитания среднего уровня фона из флуоресценции каждого образца и нормализации данных, которые затем конвертируются в логарифмическую шкалу. Далее задают пороговую линию, соответствующую 10% от образца с максимальным сигналом флуоресценции для каждого канала. Значение Ct определяется, как цикл, на котором кривая флуоресценции образца пересекает пороговую линию. Для каждого образца анализируются значения Ct по каналам для флуорофоров FAM и R6G.
Если для исследуемого образца по каналу для флуорофора FAM определено значение Ct, равное 37 или меньше, а по каналу для флуорофора R6G значение Ct отсутствует или разница между значениями Ct по каналам для флуорофоров R6G и FAM больше 2, то образец имеет генотип GG.
Если для исследуемого образца по каналу для флуорофора R6G определено Ct, равное 37 или меньше, а по каналу для флуорофора FAM значение Ct отсутствует или разница между значениями Ct по каналам для флуорофоров FAM и R6G больше 2, то образец имеет генотип АА.
Если для исследуемого образца по каналам для флуорофоров FAM и R6G определено Ct, равное или меньше 37, и разница значений Ct по модулю между каналами для флуорофоров FAM и R6G меньше 2, то образец имеет генотип GA.
Реализация заявляемого изобретения поясняется следующими примерами:
Пример 1. Получение набора олигонуклеотидных праймеров и зондов для определения rs55986091.
Для подбора последовательностей-мишеней - мест гибридизации олигонуклеотидов, используют фрагменты референсного генома, взятые из базы данных NCBI dbSNP [NCBI dbSNP https://www.ncbi.nlm.nih.gov/snp/ обращено 10.09.2021]. Для поиска гомологичных областей применяли современные алгоритмы in silico анализа нуклеотидных последовательностей и программы Primer BLAST, Unipro UGENE, MEGA.
С этой целью были проанализирован фрагмент последовательности генома, включающий rs55986091, и не имеющий гомологии с другими последовательностями ДНК. В результате проведенного анализа выбран участок гомологичной последовательности, к которому подобрали праймеры и зонды для амплификации фрагмента размером 297 пар оснований: прямой праймер 6091-F - SEQ ID NO: 1; обратный праймер 6091-R - SEQ ID NO: 2; флоуресцентный зонд 6091-G - SEQ ID NO: 3 и флоуресцентный зонд 6091-А - SEQ ID NO: 4.
Анализ заявляемых последовательностей с использованием ресурса Primer BLAST продемонстрировал, что прямой 6091-F (SEQ ID NO: 1) и обратный 6091-R (SEQ ID NO: 2) праймеры амплифицируют участок ДНК, включающий rs55986091, со 100% специфичностью. Также специфичность прямого и обратного праймеров подтверждена с помощью секвенирования нуклеотидных последовательностей.
Пример 2. Детекция rs55986091 методом ПЦР-РРВ.
ПЦР-РРВ проводили при следующих условиях:
Общий объем реакционной смеси - 25 мкл.
Компоненты ПЦР смешивали следующим образом:
(a) 10 мкл смеси, содержащей:
- олигонуклеотидные праймеры SEQ ID NO: 1, 2 - по 0,6 мкМ;
- флоуресцентный зонд SEQ ID NO: 3-0,12 мкМ;
- флоуресцентный зонд SEQ ID NO: 4-0,12 мкМ;
- dNTPs - 0,44 мМ.
(b) 0,5 мкл реактива «Полимераза TaqF» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия), содержащего рекомбинантный фермент Taq ДНК-полимеразы.
(c) 4,5 мкл ПЦР-буфера «ОТ-ПЦР-смесь-2 FEP/FRT» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия), содержащего MgCl2.
(d) выделенная ДНК - 10 мкл.
ПЦР-РРВ проводили с флуоресцентной детекцией на приборе с 5 каналами детекции - «Rotor-Gene Q» («Qiagen», Германия). Для детекции нуклеотида G в образцах биологического материала используют канал для флуорофора FAM, для нуклеотида А канал для флуорофора R6G.
Подготовленный описанным способом материал, содержащий уникальные олигонуклеотидные последовательности SEQ ID NO: 1-4 используют для определения и rs55986091 в образцах биологического материала.
Пример 3. Детекция rs55986091 в образцах ДНК, экстрагированных из цельной венозной крови.
Для детекции rs55986091 использована случайная выборка 138 человек. ПЦР-исследование проводили в соответствии с МУ 1.3.2569-09 «Организация работ лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для экстракции ДНК использовали набор реагентов «РИБО-преп» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия), концентрация ДНК - 105 копий в 10 мкл.
ПЦР-РРВ проводили при условиях, указанных в Примере 2.
Амплификацию проводили по следующей программе: 1 цикл 95°С в течение 15 минут, 45 циклов при температуре 95°С - 10 секунд / 60°С - 20 секунд / 72°С - 20 секунд. Детекция флуоресценции проводили на этапе 60°С по каналу для флуорофора БАМ - для аллеля G, по каналу для флуорофора R6G - для аллеля А.
Для анализа результатов провели пороговую линию, соответствующую величине 10% (0,07 ОЕ) от образца с максимальным сигналом флуоресценции по каналу Green для флуорофора БАМ (0,7 ОЕ) и 10% (0,12 ОЕ) от образца с максимальным сигналом флуоресценции по каналу Yellow для флуорофора R6G (1,2 ОЕ).
Разница в значении пороговых циклов по каналам для флуорофоров FAM и R6G была рассчитана для каждого образца. Если для исследуемого образца по каналу для флуорофора FAM определено значение Ct, равное 37 или меньше, а по каналу для флуорофора R6G значение Ct отсутствует или разница между значениями Ct по каналам для флуорофоров R6G и FAM больше 2, то образец имеет генотип GG.
Если для исследуемого образца по каналу для флуорофора R6G определено Ct, равное 37 или меньше, а по каналу для флуорофора FAM значение Ct отсутствует или разница между значениями Ct по каналам для флуорофоров FAM и R6G больше 2, то образец имеет генотип АА.
Если для исследуемого образца по каналам для флуорофоров FAM и R6G определено Ct, равное или меньше 37, и разница значений Ct по модулю между каналами для флуорофоров FAM и R6G меньше 2, то образец имеет генотип GA.
В результате анализа образцы были интерпретированы, как 113/138 человек с генотипом GG, 7/138 с генотипом GA и 18/138 с генотипом АА. Частота аллеля А - 88%, G - 12%. Сравнение результатов с базой данных Ensembl [Ensemble Genome Browser: https://www.ensembl.org/], не показало значимых отличий от Европейской популяции (EUR) р-значение для χ2>0,1. Полученные результаты были подтверждены секвенированием по Сэнгеру и пиросеквенированием. Отсутствие статистически значимых отличий от Европейской выборки и результаты секвенирования позволяют сделать вывод о специфичности заявляемого набора олигонуклеотидных праймеров и зондов.
Пример 4. Детекция rs55986091 в образцах ДНК, экстрагированных из соскобов со слизистой оболочки цервикального канала.
Для детекции rs55986091 использованы выборка 218 образцов ДНК женщин с отрицательными результатами выявления ВПЧ и негативными результатами в отношении интраэпителиального поражения или злокачественности при проведении цервикального скрининга.
Выделение ДНК и ПЦР-РРВ проводили, как показано в Примере 2, с использованием набора уникальных олигонуклеотидных последовательностей и зондов SEQ ID NO: 1-4.
Амплификацию и детекцию, а также оценку полученного результата проводили по программе и алгоритму, описанному в Примере 3.
В результате анализа образцы были интерпретированы, как 165/218 женщин с генотипом GG, 16/218 с генотипом GA и 37 с генотипом АА. Частота аллеля А - 79%, G - 21%. Полученные результаты были подтверждены секвенированием по Сэнгеру и пиросеквенированием. Сравнение результатов с образцами не показало значимых отличий р-значение для χ2>0,1.
Пример 5. Детекция rs55986091 в образцах ДНК женщин с РШМ, экстрагированных из соскобов со слизистой оболочки цервикального канала.
Для детекции rs55986091 была использована выборка 124 женщин с положительными результатами выявления ВПЧ и гистологически подтвержденным РШМ.
Выделение ДНК и ПЦР-РРВ проводили, как показано в Примере 2, с использованием набора уникальных олигонуклеотидных последовательностей и зондов SEQ ID NO: 1-4.
Амплификацию и детекцию, а также оценку полученного результата проводили по программе и алгоритму, описанному в Примере 3.
В результате анализа образцы были интерпретированы, как 115/124 женщин с генотипом GG, 5/124 женщин с генотипом GA и 4 женщин с генотипом АА. Частота аллеля А - 95%, G - 5%. Частота аллелей значимо отличается между выборками женщин с отрицательными анализами на ВПЧ и негативными результатами в отношении интраэпителиального поражения или злокачественности при проведении цервикального скрининга и женщин с положительными результатами выявления ВПЧ и гистологически подтвержденным РШМ.
Пример 6. Детекция rs55986091 в образцах ДНК с известным генотипом и различной концентрацией.
Для детекции rs55986091 были использованы 45 образцов с известным генотипом и различной концентрацией. Для измерения концентрации был использован набор «АмплиСенс® EBV / CMV / HHV6-скрин-FL». После измерения образцы были разведены до концентрации 2×104 копий/мл, 1×104 копий/мл и 5х103 коп/мл. Для всех образцов было проведено секвенирование по Сэнгеру.
Выделение ДНК и ПЦР-РРВ проводили, как показано в Примере 2, с использованием набора уникальных олигонуклеотидных последовательностей и зондов SEQ ID NO: 1-4.
Амплификацию и детекцию, а также оценку полученного результата проводили по программе и алгоритму, описанному в Примере 3.
В результате не было найдено разницы не по одному по образцу в сравнении с результатами секвенирования. Результаты генотипирования отражены в Таблице 2.
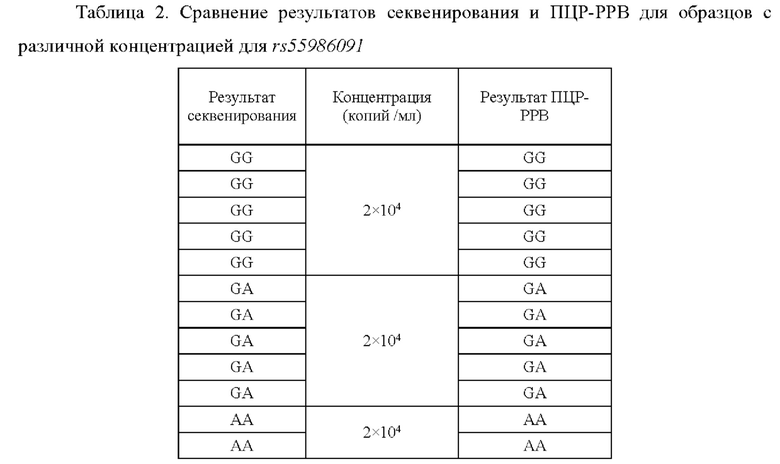
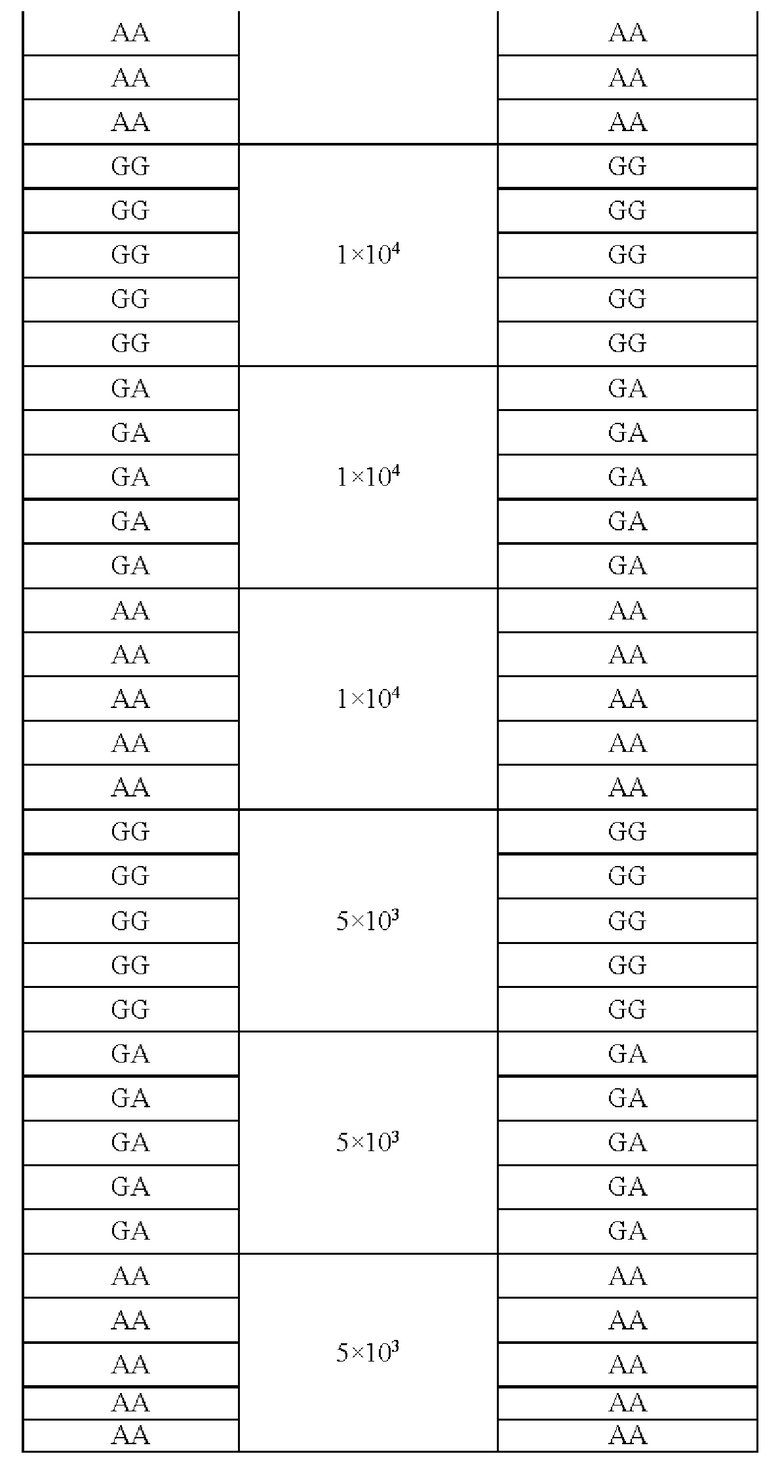
Таким образом, заявляемое изобретение позволяет детектировать аллели rs55986091 в образцах биологического материала. Набор синтезированных олигонуклеотидных праймеров и зондов SEQ ID NO: 1-4 не дает перекрестных реакций с другими последовательностями генома, амлифицирует последовательность, включающую rs55986091, со 100% специфичностью и позволяют определять наличие или отсутствие предрасположенности к раку шейки матки.
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ генотипирования гена TLR2 по полиморфизму rs5743708 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805861C1 |
| Способ генотипирования гена TLR4 по полиморфизму rs4986790 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805862C1 |
| Способ генотипирования гена TLR2 по полиморфизму rs3804100 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805860C1 |
| Способ генотипирования гена TLR8 по полиморфизму rs3764880 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805864C1 |
| Способ генотипирования гена TLR1 по полиморфизму rs5743551 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805859C1 |
| Способ генотипирования гена TLR6 по полиморфизму rs5743810 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805863C1 |
| Мультиплексная ПЦР-смесь для определения серотипов 12FAB, 15BC, 22FA, 8 Streptococcus pneumoniae и способ ее применения | 2021 |
|
RU2787181C1 |
| Олигонуклеотиды для определения 15BC серотипа Streptococcus pneumoniae | 2021 |
|
RU2795316C1 |
| Олигонуклеотиды для определения 12FAB серотипа Streptococcus pneumoniae | 2021 |
|
RU2795021C1 |
| Набор олигонуклеотидных праймеров и зондов для видовой дифференциации вируса герпеса человека 6А и вируса герпеса человека 6В и способ его применения | 2022 |
|
RU2806427C1 |
Изобретение относится к биотехнологии. Предложен набор олигонуклеотидных праймеров и зондов для определения аллей полиморфизма rs55986091, который ассоциирован с предрасположенностью к раку шейки матки. Набор включает прямой праймер 6091-F - SEQ ID NO: 1; TCT TTT TTA TTT TTC СТА AGA GTC GAT, обратный праймер 6091-R - SEQ ID NO: 2; GCA ATT TTA ATT TCG CCT CAG TC, флоуресцентный зонд 6091-G- SEQ ID NO: 3. (FAM)TTA TTA GGG ATT TTC A (BHQ1), флоуресцентный зонд 6091-А - SEHQ ID NO: 4. (R6G)TTA TTA GAG ATT TTC AA (BHQ1). Предложен способ применения набора олигонуклеотидных праймеров и зондов для определения аллей полиморфизма rs55986091. Способ предусматривает экстракцию ДНК из биологического материала и проведение ПЦР-РРВ, амплификацию, анализ и интерпретацию результатов. Наличие в образце биологического материала нукдеотида G определяется посредством оценки кривых накопления флуоресцентных сигналов по каналу для флуорофора FAM. Наличие в образце биологического материала нукдеотида А определяется посредством оценки кривых накопления флуоресцентных сигналов по каналу для флуорофора R6G. Полученный результат интерпретируют следующим образом: (i) образец биологического материала имеет генотип GG при условии, если при его исследовании по каналу для флуорофора FAM определено значение Ct, равное 37 или меньше, а по каналу для флуорофора R6G значение Ct отсутствует или разница между значениями Ct по каналам для флуорофоров R6G и FAM больше 2; (ii) образец биологического материала имеет генотип АА, если при его исследовании по каналу для флуорофора R6G определено Ct, равное 37 или меньше, а по каналу для флуорофора FAM значение Ct отсутствует или разница между значениями Ct по каналам для флуорофоров FAM и R6G больше 2; (iii) образец биологического материала имеет генотип GA, если при его исследовании каналам для флуорофоров FAM и R6G определено Ct, равное или меньше 37, и разница значений Ct по модулю между каналами для флуорофоров FAM и R6G меньше 2. Синтезированные олигонуклеотидные праймеры SEQ ID NO: 1-2 позволяют амплифицировать фрагмент между генами HLA-DQA1 и HLA-DQB1 и не дают перекрестных реакций с другими последовательностями генома; синтезированные олигонуклеотидные зонды SEQ ID NO: 3-4 позволяют определять аллели полиморфизма rs55986091 (замена G>A) и детектировать генотип со 100% специфичностью. 2 н. и 1 з.п. ф-лы, 2 табл., 6 пр.
1. Набор олигонуклеотидных праймеров и зондов для определения аллей полиморфизма rs55986091, имеющий следующий нуклеотидиый состав:
прямой праймер 6091-F - SEQ ID NO: 1; TCT TTT TTA TTT TTC СТА AGA GTC GAT,
обратный праймер 6091-R - SEQ ID NO: 2; GCA ATT TTA ATT TCG CCT CAG TC,
флоуресцентный зонд 6091-G- SEQ ID NO: 3. (FAM)TTA TTA GGG ATT TTC A (BHQ1),
флоуресцентный зонд 6091-А - SEHQ ID NO: 4. (R6G)TTA TTA GAG ATT TTC AA (BHQ1).
2. Способ применения набора олигонуклеотидных праймеров и зондов по п. 1 для определения аллей полиморфизма rs55986091, включающий экстракцию ДНК из биологического материала и проведение ПЦР-РРВ с использованием набора олигонуклеотидных праймеров и зондов по п. 1, амплификацию, анализ и интерпретацию результатов, где наличие в образце биологического материала нукдеотида G определяется посредством оценки кривых накопления флуоресцентных сигналов по каналу для флуорофора FAM, а наличие в образце биологического материала нукдеотида А определяется посредством оценки кривых накопления флуоресцентных сигналов по каналу для флуорофора R6G, при этом полуденный результат интерпретируют следующим образом:
(i) образец биологического материала имеет генотип GG при условии, если при его исследовании по каналу для флуорофора FAM определено значение Ct, равное 37 или меньше, а по каналу для флуорофора R6G значение Ct отсутствует или разница между значениями Ct по каналам для флуорофоров R6G и FAM больше 2.
(ii) образец биологического материала имеет генотип АА, если при его исследовании по каналу для флуорофора R6G определено Ct, равное 37 или меньше, а по каналу для флуорофора FAM значение Ct отсутствует или разница между значениями Ct по каналам для флуорофоров FAM и R6G больше 2.
(iii) образец биологического материала имеет генотип GA, если при его исследовании каналам для флуорофоров FAM и R6G определено Ct, равное или меньше 37, и разница значений Ct по модулю между каналами для флуорофоров FAM и R6G меньше 2.
3. Способ по п. 2, где для анализа результатов задают пороговую линию, соответствующую 10% от образца с максимальным сигналом флуоресценции для каждого канала.
| ВИНОКУРОВ М.А | |||
| и др., Генетические полиморфизмы, ассоциированные с раком шейки матки: систематический обзор, Журнал микробиологии, эпидемиологии и иммунобиологии., 30.06.2022; 99(3) DOI: https://doi.org/10.36233/0372-9311-251 | |||
| БУКУЕВ Н | |||
| М., Эпидемиология и полиморфизм генов при раке шейки матки на модели кыргызстана, диссертация, Бишкек - 2021, |
Авторы
Даты
2023-09-26—Публикация
2022-12-28—Подача