Изобретение относится к области биотехнологии, в частности к определению аллелей гена человека TLR2 по однонуклеотидному полиморфизму rs5743708 в образах биологического материала методом полимеразной цепной реакции (ПЦР) в режиме реального времени и может применяться в исследованиях по изучению влияния однонуклеотидных полиморфизмов (SNP) в генах врожденного иммунитета на возникновение и течение мультифакторных заболеваний, а также в практической деятельности при диагностировании генетических заболеваний, обусловленных таким влиянием.
Толл-подобные рецепторы (TLR) - рецепторы врожденного иммунитета, располагающиеся как на поверхности, так и внутри клетки и распознающие патоген, проникающий в организм. TLR распознают не только экзогенные молекулярные паттерны, связанные с патогеном (PAMPs), но и эндогенные молекулы, связанные с повреждением (DAMPs), которые высвобождаются из поврежденных или умирающих клеток. Активированные TLR с помощью PAMPs или DAMPs инициируют сигнальные каскады, такие как ядерный фактор-каппа В (NFkB), пути митоген-ассоциированной протеинкиназы (MAPK) и интерферона (IFN), и способствуют секреции цитокинов и хемокинов, которые регулируют иммунные и воспалительные реакции, направленные на элиминацию патогенов.
Показана связь SNP в генах TLR с предрасположенностью к возникновению и тяжелому течению мультифакторных заболеваний. Определение аллелей SNP в генах TLR может использоваться для предупреждения тяжелого течения инфекционных заболеваний.
В исследованиях однонуклеотидных полиморфизмов, связанных с туберкулезом легких, наиболее распространены полиморфизмы гена TLR2, что доказывает связь этого рецептора с распознаванием бактерий.
Аллели риска в комплексе с другими факторами могут привести к тяжелому течению инфекционных заболеваний. Аллель А в полиморфизме rs5743708 гена TLR2 (rs5743708-G) является протективным (защитным) аллелем от развития активной формы туберкулеза, аллель rs5743708-A, наоборот, является маркером риска возникновения активной формы и тяжелого туберкулеза [А.С.Ogus, В. Yoldas, Т. Ozdemir, A. Uguz, S. Olcen, I. Keser, M. Coskun, A. Cilli, O. Yegin European Respiratory Journal 2004 23: 219-223; DOI: 10.1183/09031936.03.00061703; Varshney D, Singh S, SinhaE, Mohanty KK, Kumar S, Kumar Barik S, Patil SA, Katara P. Systematic review and meta-analysis of human Toll-like receptors genetic polymorphisms for susceptibility to tuberculosis infection. Cytokine. 2022 Apr;152:155791. doi: 10.1016/j.cyto.2021.155791. Epub 2022 Feb 11. PMID: 35158257].
Детекция такой мишени, как аллель А полиморфизма rs5743708 гена TLR2 (rs5743708-A), позволит на ранних этапах определить вероятность появления осложнений и оказывать более эффективную медицинскую помощь пациентам.
Из уровня техники известен набор для обнаружения сайтов с несколькими SNP, связанных с заболеваниями детей, где сам набор для детекции, включает 31 тип праймеров для ПЦР, а ПЦР проводят в формате мультиплекс с применением электрофореза [патент 6 CN 107287283 А дата подачи 05.06.2016, дата публикации 24.10.2017]. Несмотря на то, что данное решение позволяет определить довольно обширный перечень SNP, оно не позволяет генотипировать полиморфизм rs5743708 в режиме реального времени, а применение электрофореза в ПЦР приводит к дополнительным материальным и временным затратам.
Также известны иммунологические маркеры для адьювантной терапии меланомы применяемые для прогноза ответной реакции на иммунотерапию, особенно, например, у людей с меланомой [Заявка PCT/US2013/041448, публикация WO/2013/173644, дата международной подачи 16.05.2013, дата публикации 21.11.2013]. В некоторых вариантах осуществления SNP находятся в ITGB2, SP110, ILIB, IL23R, SLC111A1, IL12B, CCR5, TNF, ILIO, CXCL12, BTNL2, ANKRD20A4, CD 14, Р2Х7, IL8, TLR2, в том числе по полиморфизму rs5743708 A/G, и/или CD209.
Известен способ прогнозирования развития иммунного ответа на биофармацевтическое лечение или диагностическое моноклональное антитело у субъекта путем идентификации одного или нескольких полиморфизмов, таких как SNP, присутствующих в одном или нескольких генах рецептора распознавания образов (PRR), где определяют наличие полиморфизма rs5743708 гена TLR2 посредством ПЦР, проводимой в формате мультиплекс [патент US 20100311052, дата подачи 26.06.2008, дата публикации 09.12.2010].
Также определение полиморфизма rs5743708 гена TLR2 посредством ПЦР, проводимой в формате мультиплекс реализовано в способы определения функционального полиморфизма фиколина-2 для прогнозирования исхода трансплантации почки [патент ЕР 2508618, дата подачи заявки 04.04.2011, дата публикации 10.10.2012].
Из уровня техники известен способ анализа полиморфных маркеров в генах метаболизма лекарственных препаратов и генах иммунного ответа при терапии острых лейкозов у детей [патент RU 2643333, дата приоритета 05.12.2016, дата регистрации 31.01.2018], которым обеспечивается определение терминальных мутаций в генах, участвующих в метаболизме основных лекарственных препаратов при терапии ОЛ (острых лейкозов), и генах иммунного ответа. Данный способ позволяет выявлять следующие полиморфизмы генов: NR3C1 rs56149945, GRIA1 rs4958351, ASNS rs10486009, rs6971012, MTHFR rs1801133, MTRR rs1801394, SLC19A1 rs1051266, SLCOIBI rs4149056, rs 11045879, ITPA rs1127354, rs7270101, TPMT rs1800462, rs1800460, rs1142345, SLC29A1 rs324148, ABCB1 rs1045642, ABCB1 rs2032582, PTPN22 rs2476601, TLR1 rs4833095, rs5743618, TLR2 rs5743708, TLR4 rs4986790, rs4986791, IL-4 rs2243250, rs2070874, IL-10 rs1800896, IL-12b rs1860508, IL7R rs1494558, rs1494555, NOD2 rs2066844, Dectin-1 rs16910526. Набор указанных в описании праймеров используется для ПЦР для получения флюоресцентно-меченного продукта в один раунд для гибридизации на биочипе.
Упомянутые выше решения позволяют определять ген TLR2, полиморфизм rs5743708 гена TLR2 без возможности генотипирования указанного полиморфизма.
По способу прогнозирования предрасположенности к посттравматическому гонартрозу определяют генотип полиморфизма rs5743708, G2258A (Arg753Gln) гена TLR2 и в полиморфных локусах выявляют аллель А и генотипы АА и AG и при их наличии диагностируют высокую генетическую предрасположенность к развитию посттравматического гонартроза [патент RU 2770004, дата приоритета 30.07.2021, дата публикации 14.04.2022]. Генотипирование полиморфизма гена осуществляют путем выделения геномной ДНК из периферической крови и проведения, повторяющихся 35 циклов, каждый из которых включает денатурацию двухцепочечной ДНК-матрицы, отжиг праймеров и элонгацию, или синтез ДНК при заданных температурах с разделением продуктов ПЦР (ампликонов) посредством с детекцией продукта методом электрофореза в агарозном геле. Согласно изобретению, в полиморфных локусах выявляют только аллель А и генотипы АА и AG и при их наличии диагностируют высокую генетическую предрасположенность к развитию посттравматического гонартроза. Проведение ПЦР посредством электрофореза в агарозном геле приводит к увеличению временных и ресурсных издержек.
Известны наборы реагентов для определения аллелей SNP методом ПЦР в режиме реального времени. Наборы реагентов включают в себя олигонуклеотиды для детекции нескольких специфичных аллелей в исследуемых образцах и отличаются удобством для пользователя. Для выбранных мишеней детекции не всегда подтверждена связь с заболеванием и поэтому такие наборы не всегда могут использоваться в клинической практике, но используются в научно-исследовательских целях. Из уровня техники также известен набор реагентов «ThermoFisher Scientific С_30687121_20» для определения аллелей rs5743565 гена TLR1 [https://www.thermofisher.com/order/genome-database/details/genotyping/C_30687121_20?CID=&ICID=&subtype=#more-information-section]. Однако данное решение не предназначено для определения аллелей rs574708 гена TLR2.
Также генотипирование локусов rs5743708 выполняли с применением реагентов для выделения ДНК («РИБО-преп»), амплификации и пробоподготовки («ПИРО-преп») производства Центрального НИИ эпидемиологии Роспотребнадзора («АмплиСенс», Россия) методом пиросеквенирования с использованием реагентов PyroMark Q96 и системы генетического анализа PyroMark Q24 («Qiagen», Германия) [М.А. Карнаушкина, А.С.Гурьев, К.О. Миронов, Е.А. Дунаева, В.И. Корчагин, О.Ю. Бобкова, И.С. Васильева, Д.В. Кассина, М.М. Литвинова. Ассоциации полиморфизмов генов толл-подобных рецепторов и активности нетоза как прогностические критерии тяжести течения пневмонии. http://www.stm-journal.ru/ru/numbers/2021/3/1723/html]. Согласно описанному способу были определены rs5743708-A и rs5743708-G гена TLR2 методом пиросеквенирования, что делает его более затратным и трудоемким.
Таким образом, существует потребность в расширении точного диагностического инструментария, позволяющего в короткие сроки и с наименьшими затратами генотипировать аллели гена TLR2 по полиморфизму rs5743708 для своевременного и адекватного назначения диагностических, терапевтических и профилактических мероприятий, а также для дальнейшего изучения врожденного иммунитета и механизма возникновения и течения мультифакторных заболеваний.
Технический результат заявляемого изобретения направлен на генотипирование аллелей А и G гена TLR2 по полиморфизму rs5743708, а именно определение rs5743708-A и rs5743708-G гена TLR2, в образцах биологического материала ДНК с использованием широко распространенных методик и доступного оборудования.
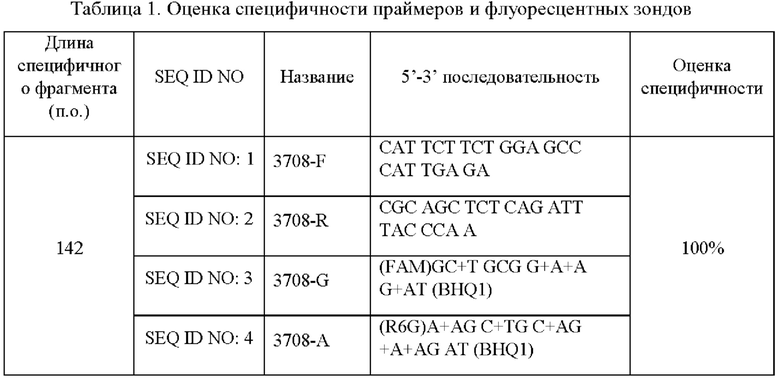
Заявленный технический результат достигается за счет проведения полимеразной цепной реакции (далее - ПЦР) с гибридизационно-флуоресцентной детекцией продуктов амплификации в режиме реального времени с применением реакционной смеси, содержащей набор уникальных синтезированных нуклеотидных праймеров и конформационно-блокированных зондов, а генотипирование осуществляют посредством оценки кривых накопления флуоресцентных сигналов по соответствующим каналам для флуорофоров, а именно: для rs5743708- G по каналу FAM, для rs5743708-A по каналу R6G.
Упомянутый набор синтезированных нуклеотидных праймеров и зондов позволяет эффективно детектировать полиморфный нуклеотид rs5743708 в последовательности ДНК и имеет следующий олигонуклеотидный состав:
прямой праймер 3708-F - SEQ ID NO: 1,
обратный праймер 3708-R - SEQ ID NO: 2,
флуоресцентный зонд 3708-G - SEQ ID NO: 3,
флуоресцентный зонд 3551-А - SEQ ID NO: 4.
Праймеры представляют собой последовательности олигонуклеотидов для амплификации фрагмента, включающей полиморфизм rs5743708, флуоресцентные зонды являются олигонуклеотидами, содержащими флуорофор, гаситель флуоресценции, позволяющими детектировать аллели в амплифицированный фрагмент.
Заявляемый набор олигонуклеотидных праймеров и зондов разработан на основе проведенного анализа последовательностей: гена TLR2 toll like receptor 2 (Homo sapiens (human)), Gene ID: 7097 NC_000004.12 GRCh38.p14 (GCF_000001405.40), представленных в базе данных National Center for Biotechnology Information (NCBI) [NCBI https://www.ncbi.nlm.nih.gov/gene/7097], и не имеющих гомологии с другими последовательностями ДНК. В результате выбран фрагмент для детекции участка, содержащего rs5743708 гена TLR2 к которому подобраны прямой и обратный праймеры для амплификации фрагмента длиной 142 пары оснований, а также флуоресцентные зонды для детекции аллелей G и А выбранного SNP: прямой праймер 3708-F - SEQ ID NO: 1; обратный праймер 3708-R - SEQ ID NO: 2; флуоресцентный зонд 3708-G - SEQ ID NO: 3; флуоресцентный зонд 3708-А - SEQ ID NO: 4.
При этом зонды содержат конформационно-блокированные нуклиотиды, которые позволяют повысить стабильность дуплекса с ДНК-мишенью. Флуоресцентный зонд 3708-G содержит 4 блокатора, которые расположены в положении 3, 8, 9, 11. Флуоресцентный зонд 3708-А содержит 5 блокаторов, которые расположены в положении 2, 5, 8, 10, 11.
Для подбора мишеней - мест посадки олигонуклеотидов используют фрагменты референсного генома, взятые из базы данных NCBI dbSNP [NCBI dbSNP https://www.ncbi.nlm.nih.gov/snp/обращено 12.12.2022]. Для поиска гомологичных областей применяют современные алгоритмы in silico анализа нуклеотидных последовательностей и программы, находящиеся в открытом доступе: Primer BLAST, Unipro UGENE. Состав набора олигонуклеотидных праймеров и зондов приведен в Таблице 1.
Анализ результатов продемонстрировал, что прямой праймер 3708-F (SEQ ID NO: 1) и обратный праймер 3708-R (SEQ ID NO: 2) амплифицируют участок гена человека TLR2, содержащий полиморфизм rs5743708 со 100% специфичностью. Также специфичность прямого и обратного праймеров подтверждена с помощью прямого метода пиросеквенирования нуклеотидных последовательностей.
где FAM флуорофор для SEQ ID NO: 3, R6G флуорофор для SEQ ID NO: 4, BHQ1 - гаситель флуоресценции, соответствующие красителю каналов флуороворов FAM и R6G, знак «+» - перед конформационно-блокированным нуклеотидом.
Заявляемое изобретение является результатом научно-исследовательской работы лаборатории молекулярных методов изучения генетических полиморфизмов «Изучение генетической предрасположенности к мультифакторным заболеваниям», проведенной ФБУН ЦНИИ Эпидемиологии Роспотребнадзора (Москва, Россия).
В качестве биологического материала предпочтительно использовать венозную кровь.
ПЦР-исследование проводят в соответствии с МУ 1.3.2569-09 «Организация работ лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для экстракции ДНК может быть использован комплект реагентов «РИБО-преп» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) или любой аналогичный набор/комплект реагентов для выделения ДНК, в соответствии с инструкцией производителя. Оптимальная концентрация ДНК - 103-105 копий в 10 мкл. ПЦР в режиме реального времени проводится с применением заявляемого набора олигонуклеотидных праймеров и зондов для детекции аллелей полиморфизма rs5743708 гена человека TLR2, представленных в Таблице 1.
ПЦР проводят при следующих условиях:
Общий объем реакционной смеси - 25 мкл.
Компоненты ПЦР смешиваются следующим образом:
(а) 10 мкл смеси, содержащей:
-олигонуклеотидные праймеры SEQ ID NO NO: 1, 2 - по 0,5 мМ;
-флуоресцентные зонды SEQ ID NO NO: 3, 4 - по 0,08 мМ
-dNTPs - 0,44 мМ.
(b) реактив, содержащий рекомбинантный фермент Taq ДНК-полимеразу, например, 0,5 мкл «Полимераза TaqF» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Москва, Россия) или любого аналогичного коммерческого набора в соответствии с инструкцией производителя.
(c) ПЦР-буфер, содержащий MgCl2, например, 5,0 мкл ПЦР-буфера «ОТ-ПЦР-смесь-2 FEP/FRT» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Москва, Россия) или любого аналогичного коммерческого набора в соответствии с инструкцией производителя.
(d) экстрагированная из цельной крови человека ДНК 10 мкл.
Амплификацию проводят на амплификаторе с возможностью детекции флуоресцентного сигнала как минимум по 2 каналам флуоресценции, например, «Rotor-Gene Q» («Qiagen», Германия) в соответствии с инструкцией производителя.
Амплификацию проводят по следующей программе: 1 цикл 95°С в течение 15 минут, 45 циклов при температуре 95°С - 10 секунд / 60°С - 20 секунд. Детекция флуоресценции проводится на этапе 60°С по каналу для флуорофора FAM - для rs5743708-G, по каналу для флуорофора R6G - для rs5743708-A
Анализ данных проводится на основе детекции амплификатором уровня флуоресцентного сигнала. Результаты интерпретируются на основании наличия (или отсутствия) пересечения кривой флуоресценции S-образной (сигмообразной) формы с установленной на соответствующем уровне пороговой линией. Это определяет наличие или отсутствие для анализируемого образца значения порогового цикла. В процессе амплификации ДНК зонды конкурируют за связывание с матрицей ДНК, содержащем два праймера SEQ ID NO: 1 и SEQ ID NO: 2. При этом преимущество получает зонд, соответствующий представленному в образце аллели. Если присутствуют два аллеля полиморфизма, гибридизуются оба зонда.
Для каждого канала задают пороговую линию, соответствующую величине 10% от максимального сигнала флуоресценции образца среди образцов с определенным аллелем. Образцы, для которых кривые флуоресценции пересекают пороговую линию по каналу для детекции флуорофора FAM и при этом кинетика накопления флуоресцентного сигнала является экспоненциальной, содержат аллель G полиморфизма rs5743708 гена TLR2. Образцы, для которых кривые флуоресценции пересекают пороговую линию по каналу для детекции флуорофора R6G и при этом кинетика накопления флуоресцентного сигнала является экспоненциальной, содержат аллель А полиморфизма rs5743708 гена TLR2.
Образцы, для которых кривые флуоресценции пересекли пороговые линии по каналам для детекции флуорофоров FAM и R6G и при этом кинетика накопления флуоресцентного сигнала является экспоненциальной, содержат rs5743708-G и rs5743708-А.
Реализация заявляемого изобретения поясняется следующими примерами:
Пример 1. Получение набора олигонуклеотидных праймеров и зондов для генотипирования аллелей гена TLR2 по полиморфизму rs5743708
Для подбора целевых последовательностей - мест посадки олигонуклеотидов, использовали фрагменты гена TLR2 toll like receptor 2 (Homo sapiens (human)), Gene ID: 7097 NC_000004.12 GRCh38.p14 (GCF_000001405.40), представленных в базе данных National Center for Biotechnology Information (NCBI) [NCBI https://www.ncbi.nlm.nih.gov/gene/7097], и не имеющих гомологии с другими последовательностями ДНК. Для поиска гомологичных областей использовали современные алгоритмы in silico анализа нуклеотидных последовательностей и программы Primer BLAST, Unipro UGENE, MEGA.
В результате проведенного анализа выбран участок гомологичной последовательности, к которому подобрали праймеры и зонды для амплификации фрагмента длиной 142 пар оснований, а также флуоресцентные зонды для детекции аллелей G и А выбранного SNP: прямой праймер 3708-F - SEQ ID NO: 1; обратный праймер 3708-R - SEQ ID NO: 2; флуоресцентный зонд 3708-G - SEQ ID NO: 3; флуоресцентный зонд 3708-А - SEQ ID NO: 4. При этом каждый зонд содержат конформационно заблокированные нуклиотиды, а именно: флуоресцентный зонд 3708-G содержит 4 блокатора, которые расположены в положении 3, 8, 9, 11. Флуоресцентный зонд 3708-А содержит 5 блокаторов, которые расположены в положении 2, 5, 8, 10, 11.
Анализ упомянутых последовательностей с использованием ресурса Primer BLAST [https://www.ncbi.nlm.nih.gov/tools/primer-blast/] показал, что прямой 3708-F и обратный 3708-R праймеры амплифицируют участок, содержащий полиморфизм rs5743708 в гене TLR2 со 100% специфичностью.
Пример 2. Детекция аллелей G и А гена TLR2 по полиморфизму rs5743708 методом ПЦР в режиме реального времени.
ПЦР в режиме реального времени проводили при следующих условиях.
Общий объем реакционной смеси - 25 мкл.
Компоненты ПЦР смешиваются следующим образом:
(а) 10 мкл смеси, содержащей:
- олигонуклеотидные праймеры: SEQ ID NO: 1, SEQ ID NO: 2 - по 0,5 мМ;
- флуоресцентные зонды SEQ ID NO: 3, SEQ ID NO: 4 - по 0,008 мМ;
- dNTPs - 0,44 мМ.
(b) 0,5 мкл реактива «Полимераза TaqF» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия), содержащего рекомбинантный фермент Taq ДНК-полимеразу.
(c) 5,0 мкл ПЦР-буфера «ОТ-ПЦР-смесь-2 FEP/FRT» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия), содержащего MgCl2;
(d) экстрагированная из цельной венозной крови человека ДНК - 10 мкл.
ПЦР в режиме реального времени проводили с флуоресцентной детекцией результата на приборе с 5 каналами детекции - «Rotor-Gene Q» («Qiagen», Германия).
Подготовленный описанным способом материал, содержащий уникальные олигонуклеотидные последовательности SEQ ID NO: 1-4, использовали для определения аллелей rs5743708 гена TLR2 в образцах биологического материала.
Пример 3. Генотипирование аллелей G и А гена TLR2 по полиморфизму rs5743708 в образцах биологического материала.
Для определения аллелей rs5743708 гена человека TLR2 в образцах биологического материала выбрано 60 проб, для исследования использовали биологический материал - венозная кровь. Выделение ДНК проводили в соответствии с МУ 1.3.2569-09 «Организация работ лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для экстракции ДНК использовали набор реагентов «РИБО-преп» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) в соответствии с инструкцией производителя.
ПЦР-РРВ проводили в условиях, описанных в Примере 2, с использованием уникальных синтезированных олигонуклеотидных праймеров и зондов SEQ ID NO: 1-4.
Амплификацию проводили на приборе «Rotor-Gene Q» («Qiagen», Германия) по следующей программе: 1 цикл 95°С в течение 15 минут, 45 циклов при температуре 95°С - 10 секунд / 60°С - 20 секунд. Детекция флуоресцентного сигнала проводилась на этапе 60°С по каналу для флуорофора FAM - для алелля G, и по каналу для флуорофора R6G - для алелля А.
При анализе результатов для каждого канала задали пороговую линию, соответствующую величине 10% от максимального сигнала флуоресценции образца среди образцов с определенным аллелем.
Для 37/60 образцов кривая флуоресценции пересекла пороговую линию по каналу для флуорофора FAM, для 23/60 образцов кривая флуоресценции пересекла пороговую линию по каналу для флуорофора R6G, при этом кинетика накопления флуоресцентных сигналов была экспоненциальной.
По каналу для флуорофора FAM определили наличие аллеля rs5743708-G, по каналу для флуорофора R6G определили наличие аллеля rs5743708-A полиморфизма.
Результаты исследования были подтверждены с помощью метода пиросеквенирования нуклеотидных последовательностей, которое выполнялось посредством системы генетического анализа PyroMark Q24 («Qiagen», Германия) в соответствии с инструкцией производителя.
Пример 4 Генотипирование аллелей G и А гена TLR2 по полиморфизму rs5743708 в образцах биологического материала.
Для определения аллелей rs5743708 гена человека TLR2 в образцах биологического материала выбрано 17 проб, для исследования использовали биологический материал - цельная кровь. Выделение ДНК проводили в соответствии с МУ 1.3.2569-09 «Организация работ лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для экстракции ДНК использовали набор реагентов «РИБО-преп» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) в соответствии с инструкцией производителя.
ПЦР-РРВ проводили в условиях, описанных в Примере 2, с использованием уникальных синтезированных олигонуклеотидных праймеров и зондов SEQ ID NO: 1-4.
Амплификацию проводили на приборе «Rotor-Gene Q» («Qiagen», Германия) по следующей программе: 1 цикл 95°С в течение 15 минут, 45 циклов при температуре 95°С - 10 секунд / 60°С - 20 секунд. Детекция флуоресцентного сигнала проводилась на этапе 60°С по каналу для флуорофора FAM для rs5743708-G, и по каналу для флуорофора R6G для rs57437081-A.
При анализе результатов для каждого канала задали пороговую линию, соответствующую величине 10% от максимального сигнала флуоресценции образца среди образцов с определенным аллелем.
Для 17/17 образцов кривая флуоресценции пересекла пороговую линию по каналу для флуорофора FAM, что позволило определить наличие аллеля G полиморфизма rs5743708 гена TLR2 в 17 образцах из выборки.
Результаты исследования были подтверждены с помощью метода пиросеквенирования нуклеотидных последовательностей, которое выполнялось посредством системы генетического анализа PyroMark Q24 («Qiagen», Германия) в соответствии с инструкцией производителя.
Пример 5. Генотипирование аллелей G и А гена TLR2 по полиморфизму rs5743708 в образцах биологического материала.
Для определения аллелей rs5743708 гена человека TLR2 в образцах биологического материала выбрано 8 проб, для исследования использовали биологический материал - венозная кровь. Выделение ДНК проводили в соответствии с МУ 1.3.2569-09 «Организация работ лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для экстракции ДНК использовали набор реагентов «РИБО-преп» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) в соответствии с инструкцией производителя.
ПЦР-РРВ проводили в условиях, описанных в Примере 2, с использованием уникальных синтезированных олигонуклеотидных праймеров и зондов SEQ ID NO: 1-4.
Амплификацию проводили на приборе «Rotor-Gene Q» («Qiagen», Германия) по следующей программе: 1 цикл 95°С в течение 15 минут, 45 циклов при температуре 95°С - 10 секунд / 60°С - 20 секунд. Детекция флуоресцентного сигнала проводилась на этапе 60°С по каналу для флуорофора FAM для rs5743708-G, и по каналу для флуорофора R6G для rs5743708-A.
При анализе результатов для каждого канала задали пороговую линию, соответствующую величине 10% от максимального сигнала флуоресценции образца среди образцов с определенным аллелем.
Для 8/8 образцов кривая флуоресценции пересекла пороговую линию по каналу для флуорофора R6G, что позволило определить наличие аллеля А полиморфизма rs5743708 гена TLR2 в 8 образцах из выборки.
Результаты исследования были подтверждены с помощью метода пиросеквенирования нуклеотидных последовательностей, которое выполнялось посредством системы генетического анализа PyroMark Q24 («Qiagen», Германия) в соответствии с инструкцией производителя.
Пример 6. Генотипирование аллелей G и А гена TLR2 по полиморфизму rs5743708 в образцах биологического материала.
Для определения аллелей rs5743708 гена человека TLR2 в образцах биологического материала выбрано 56 проб, для исследования использовали биологический материал - цельная кровь. Выделение ДНК проводили в соответствии с МУ 1.3.2569-09 «Организация работ лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для экстракции ДНК использовали набор реагентов «РИБО-преп» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) в соответствии с инструкцией производителя.
ПЦР в режиме реального времени проводили в условиях, описанных в Примере 2, с использованием уникальных синтезированных олигонуклеотидных праймеров и зондов SEQ ID NO: 1-4.
Амплификацию проводили на приборе «Rotor-Gene Q» («Qiagen», Германия) по следующей программе: 1 цикл 95°С в течение 15 минут, 45 циклов при температуре 95°С - 10 секунд / 60°С - 20 секунд. Детекция флуоресцентного сигнала проводилась на этапе 60°С по каналу для флуорофора FAM для алелля G, и по каналу для флуорофора R6G для алелля А.
При анализе результатов для каждого канала задали пороговую линию, соответствующую величине 10% от максимального сигнала флуоресценции образца среди образцов с определенным аллелем.
Для 21/56 образцов кривая флуоресценции пересекла пороговую линию по каналу для флуорофора FAM, для 18/56 образцов кривые флуоресценции пересекли пороговую линию по каналу для флуорофора R6G, для 17/56 образцов кривая флуоресценции пересекла пороговые линии по каналам для флуорофора FAM и R6G, при этом кинетика накопления флуоресцентных сигналов была экспоненциальной.
По каналу для флуорофора FAM определили наличие ДНК rs5743708-G, по каналу для флуорофора R6G определили наличие ДНК rs5743708-A, наличие сигналов по двум каналам для флуорофоров FAM и R6G одновременно подтвердили присутствие ДНК двух аллелей - rs5743708-G и rs5743708-A.
Результаты исследования были подтверждены с помощью метода пиросеквенирования нуклеотидных последовательностей, которое выполнялось посредством системы генетического анализа PyroMark Q24 («Qiagen», Германия) в соответствии с инструкцией производителя.
Таким образом, заявляемое изобретение позволяет детектировать аллели rs5743708-G и rs5743708-A гена TLR2 в образцах биологического материала. Набор уникальных синтезированных олигонуклеотидных праймеров и зондов SEQ ID NO: 1-4 не дает перекрестных реакций с другими последовательностями генома, амплифицирует последовательность, включающую rs5743708, со 100% специфичностью и позволяют однозначная интерпретировать полученные результаты.
--->
<ST26SequenceListing dtdVersion="V1_2" fileName="rs5743708"
softwareName="WIPO Sequence" softwareVersion="1.0.0"
productionDate="2023-04-12">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>no</ApplicationNumberText>
<FilingDate>2023-04-11</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>01</ApplicantFileReference>
<ApplicantName languageCode="ru">ФБУН ЦНИИ Эпидемиологии
Роспотребнадзора</ApplicantName>
<ApplicantNameLatin>FBUN CRIE</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ генотипирования гена TLR2 по
полиморфизму rs5743708 и набор олигонуклеотидных праймеров и зондов
для его реализации</InventionTitle>
<SequenceTotalQuantity>4</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>rs5743708</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cattcttctggagcccattgaga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>rs5743708</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgcagctctcagatttacccaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>12</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..12</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>rs5743708</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctgcggaagat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>14</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..14</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>rs5743708</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aagctgcagaagat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ генотипирования гена TLR4 по полиморфизму rs4986790 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805862C1 |
| Способ генотипирования гена TLR2 по полиморфизму rs3804100 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805860C1 |
| Способ генотипирования гена TLR6 по полиморфизму rs5743810 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805863C1 |
| Способ генотипирования гена TLR8 по полиморфизму rs3764880 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805864C1 |
| Способ генотипирования гена TLR1 по полиморфизму rs5743551 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805859C1 |
| Набор олигонуклеотидных праймеров и зондов для определения аллей полиморфизма rs55986091 и способ его применения | 2022 |
|
RU2804110C1 |
| Набор олигонуклеотидных праймеров и зондов для видовой дифференциации вируса герпеса человека 6А и вируса герпеса человека 6В и способ его применения | 2022 |
|
RU2806427C1 |
| Мультиплексная ПЦР-смесь для определения серотипов 12FAB, 15BC, 22FA, 8 Streptococcus pneumoniae и способ ее применения | 2021 |
|
RU2787181C1 |
| Способ генотипирования однонуклеотидного варианта rs2304277 (G>A) гена OGG1 человека методом полимеразно-цепной реакции в режиме реального времени | 2024 |
|
RU2836047C1 |
| Способ генотипирования однонуклеотидного варианта rs1052133 (C>G) гена OGG1 человека методом полимеразно-цепной реакции в режиме реального времени | 2024 |
|
RU2835399C1 |
Изобретение относится к биотехнологии. Предложен способ генотипирования аллелей G и А гена TLR2 по полиморфизму rs5743708, включающий: экстракцию ДНК из биологического материала, проведение ПЦР в режиме реального времени с применением реакционной смеси, содержащей набор нуклеотидных праймеров и зондов SEQ ID NO: 1-4, амплификацию, анализ и интерпретацию результатов, где наличие аллеля G гена TLR2 по полиморфизму rs5743708 определяется посредством оценки кривых накопления флуоресцентных сигналов по каналу для флуорофора FAM, а наличие в образце биологического материала аллели А гена TLR2 по полиморфизму rs5743708 определяется посредством оценки кривых накопления флуоресцентных сигналов по каналу для флуорофора R6G. Предложен набор олигонуклеотидных праймеров и зондов для применения в способе генотипирования. Изобретение позволяет детектировать аллели rs5743708-G и rs5743708-A гена TLR2 в образах биологического материала методом ПЦР в режиме реального времени. 2 н. и 3 з.п. ф-лы, 1 табл., 6 пр.
1. Способ генотипирования аллелей G и А гена TLR2 по полиморфизму rs5743708, включающий: экстракцию ДНК из биологического материала, проведение ПЦР в режиме реального времени с применением реакционной смеси, содержащей набор нуклеотидных праймеров и зондов SEQ ID NO: 1-4, амплификацию, анализ и интерпретацию результатов, где наличие аллеля G гена TLR2 по полиморфизму rs5743708 определяется посредством оценки кривых накопления флуоресцентных сигналов по каналу для флуорофора FAM, а наличие в образце биологического материала аллели А гена TLR2 по полиморфизму rs5743708 определяется посредством оценки кривых накопления флуоресцентных сигналов по каналу для флуорофора R6G.
2. Способ генотипирования но п. 1, где наличие в образце биологического материала аллеля G гена TLR2 по полиморфизму rs5743708 и аллеля А гена TLR2 по полиморфизму rs5743708 определяется посредством оценки кривых накопления флуоресцентных сигналов по каналам FAM и R6G одновременно.
3. Способ применения набора олигонуклеотидных праймеров и зондов по пп. 1 и 2, где для анализа результатов задают пороговую линию, соответствующую величине 10% от максимального сигнала флуоресценции образца среди образцов с определенным аллелем.
4. Набор олигонуклеотидных праймеров и зондов для применения в способе генотипирования по п. 1, имеющий следующий нуклеотидный состав:
прямой праймер 3708-F - SEQ ID NO: 1;
обратный праймер 3708-R - SEQ ID NO: 2;
флуоресцентный зонд 3708-G - SEQ ID NO: 3;
флуоресцентный зонд 3708-A - SEQ ID NO: 4,
при этом флуоресцентный зонд 3708-G SEQ ID NO: 3 содержит: конформационно-блокированные нуклеотиды, расположенные в положении 3, 8, 9, 11, а флуоресцентный зонд 3708-А SEQ ID NO: 4 содержит конформационно-блокированные нуклеотиды, расположенные в положении 2, 5, 8, 10, 11.
5. Набор олигонуклеотидных праймеров и зондов по п. 4, где для создания прямого и обратного праймеров, флуоресцентных зондов используют фрагмент гена TLR2 Homo sapiens.
| Способ прогнозирования предрасположенности к посттравматическому гонартрозу | 2021 |
|
RU2770004C1 |
| Способ анализа полиморфных маркеров в генах метаболизма лекарственных препаратов и генах иммунного ответа при терапии острых лейкозов у детей | 2016 |
|
RU2643333C1 |
| US 20100311052 A1, 09.12.2010 | |||
| М.А | |||
| Карнаушкина и др | |||
| Ассоциации полиморфизмов генов толл-подобных рецепторов и активности нетоза как прогностические критерии тяжести течения пневмонии, том 13, номер 3 (2021), весь документ, http://www.stm-journal.ru/ru/numbers/2021/3/1723/html | |||
| Барбараш О.Л | |||
Авторы
Даты
2023-10-24—Публикация
2023-04-13—Подача