Область техники
Изобретение относится к области биотехнологии и молекулярной биологии, в частности генной инженерии, а именно к способу получения рекомбинантных частиц вируса везикулярного стоматита, кодирующих белок интереса.
Уровень техники
Высокий терапевтический потенциал онколитических вирусов раскрылся в последние 2 года в ходе исследований эффекта их применения в комбинации с различными ингибиторами контрольных точек иммунного ответа (immune checkpoint inhibitors) при лечении резистентных, неоперабельных или рецидивирующих форм рака [Russell L. et al. Oncolytic viruses: priming time for cancer immunotherapy // BioDrugs. - 2019. - T. 33. - №. 5. - C. 485-501]. Например, эффективность терапии значительно возрастала при использовании препарата Imlygic® (talimogene laherparepvec) в комбинации с пембролизумабом, ниволумабом и ипилимумабом [Dummer R, Hoeller С, Gruter IP, Michielin О. Combining talimogene laherparepvec with immunotherapies in melanoma and other solid tumors. Cancer Immunol Immunother. 2017 Jun;66(6):683-695. doi: 10.1007/s00262-017-1967-1]. В настоящее время разработка онколитических вирусов является одним из приоритетных направлений иммунотерапии опухолей. Ведется более 100 клинических исследований ранних фаз [Pearl Т.М. et al. Oncolytic virus-based cytokine expression to improve immune activity in brain and solid tumors // Molecular Therapy-Oncolytics. - 2019. - T. 13. - C. 14-21; Zheng M. et al. Oncolytic viruses for cancer therapy: barriers and recent advances // Molecular Therapy-Oncolytics. - 2019. - T. 15. - C. 234-247].
Вирус везикулярного стоматита (VSV, везикуловирус, везикуловирус Индианы) является перспективным для создания на его основе безопасного и эффективного онколитика, в особенности в комбинации с иммунотерапевтическими подходами. Низкая патогенность (вирус поражает крупный домашний скот, а в случае заражения человека вирус чаще всего не вызывает никаких симптомов), репликация в цитоплазме (без интеграции в геном), возможность получения высокого вирусного титра при наработке VSV в культурах клеток млекопитающих (BHK21, HEK293) и простота выделения, легкость генетической модификации, отсутствие в популяции нейтрализующих антител сделали VSV идеальным кандидатом для получения вирусных вакцин и онколитических вирусов [Geisbert Т.W., Feldmann Н. Recombinant vesicular stomatitis virus-based vaccines against Ebola and Marburg virus infections // The Journal of infectious diseases. - 2011. - T. 204. - №. suppl_3. - C. S1075-S1081; Zemp F., Rajwani J., Mahoney D.J. Rhabdoviruses as vaccine platforms for infectious disease and cancer // Biotechnology and Genetic Engineering Reviews. - 2018. - T. 34. №. 1. - C. 122-138.; Velazquez-Salinas L. et al. Oncolytic recombinant vesicular stomatitis virus (VSV) is nonpathogenic and nontransmissible in pigs, a natural host of VSV // Human Gene Therapy Clinical Development. - 2017. - T. 28. - №. 2. - C. 108-115].
VSV представляет собой вирус с отрицательной цепью РНК семейства Rhabdoviridae, в геноме которого содержатся 5 генов, кодирующих структурные вирусные белки:
1) N (nucleocapsid protein) отвечает за упаковку вирусного генома, а также играет важную роль при транскрипции и репликации;
2) Р (phosphoprotein) обеспечивает связь между полимеразой и нуклеокапсидом и является регулятором активности и конформации полимеразы;
3) М (matrix protein) отвечает за сборку вирионов, а также подавляет противовирусный интерфероновый сигналинг, ингибируя экспорт клеточной мРНК через прямое взаимодействие с комплексом RAE1-NUP98;
4) G (glycoprotein) поверхностный белок вируса, который обеспечивает эндоцитоз вирусных частиц через взаимодействие с LDL рецепторам, что объясняет широкий тропизм VSV;
5) L (RNA-dependent RNA polymerase) полифункциональный фермент, обеспечивающий транскрипцию и репликацию, кэпирование, метилирование и полиаденилирование [Rose J., Schubert М. Rhabdovirus genomes and their products // The rhabdoviruses. - Springer, Boston, MA, 1987. - C. 129-166.; Faria P.A. et al. VSV disrupts the Rael/mrnp41 mRNA nuclear export pathway // Molecular cell. - 2005. - T. 17. - №. 1. - C. 93-102; Jenni S. et al. Structure of the vesicular stomatitis virus L protein in complex with its phosphoprotein cofactor // Cell reports. - 2020. - T. 30. - №. 1. - C. 53-60. e5.; Rahmeh A.A. et al. Critical phosphoprotein elements that regulate polymerase architecture and function in vesicular stomatitis virus // Proceedings of the National Academy of Sciences. - 2012. - T. 109. - №. 36. - C. 14628-14633.; Morin В., Whelan S.P.J. Sensitivity of the polymerase of vesicular stomatitis virus to 2' substitutions in the template and nucleotide triphosphate during initiation and elongation // Journal of Biological Chemistry. - 2014. - T. 289. - №. 14. - C. 9961-9969].
В здоровых клетках человека вирус не может воспроизводиться из-за интерферонового ответа, который позволяет клеткам адекватно реагировать на вирусную инфекцию. Этого нельзя сказать о раковых клетках, не реагирующих на интерферон, - это позволяет VSV преимущественно расти в раковых клетках и лизировать их [Stojdl DF, Lichty В, Knowles S, Marius R, Atkins H, Sonenberg N, Bell JC. Exploiting tumor-specific defects in the interferon pathway with a previously unknown oncolytic virus. Nat Med. 2000 Jul;6(7):821-5. doi: 10.1038/77558].
Сборка рекомбинантного репликативно-дефектного rVSV-dG-GFP, несущего ген GFP между генами G и L, или, например, репликативно-активного VSV (rVSV-GFP) по классическому протоколу [Whitt MA. Generation of VSV pseudotypes using recombinant AG-VSV for studies on virus entry, identification of entry inhibitors, and immune responses to vaccines. J Virol Methods. 2010 Nov;169(2):365-74. doi: 10.1016/j.jviromet.2010.08.006], на который ориентируется большинство исследователей, работающих с VSV, включает трансфекцию клеток BHK21 шестью различными плазмидами (пятью в случае с репликативно активным VSV), которые экспрессируют антигеномно-смысловую (или положительно-смысловую) РНК VSV, кДНК белков N, Р, G (не используется в случае наличия G гена в составе корового вектора для репликативно активного VSV), L VSV, которые находятся под контролем промотора Т7, а также включает ко-инфекцию клеток на первом этапе вирусом осповакцины (Vaccinia virus) или иным вирусом, экспрессирующим Т7 полимеразу, необходимую для сборки VSV.
В работах [Whitt MA. Generation of VSV pseudotypes using recombinant ΔG-VSV for studies on virus entry, identification of entry inhibitors, and immune responses to vaccines. J Virol Methods. 2010 Nov;169(2):365-74; Кононова A.A. Псевдовирусная система на основе вируса везикулярного стоматита для поиска противовирусных средств, действующих на вирусные поверхностные белки. Диссертация на соискание ученой степени кандидата биологических наук, Новосибирск, 2020, защита состоялась 18.09.2020 г.] описан способ получения вирусных частиц псевдотипа rVSV-ΔG с ограниченной репликацией в клеточных культурах. Процесс состоит из нескольких последовательных стадий (Фиг. 1). Клетки линии BHK21 (baby hamster kidney) заражают рекомбинантным вирусом осповакцины (Vaccinia virus), экспрессирующей РНК-полимеразу бактериофага Т7 (vvT7) и затем трансфицируют пятью плазмидами:
плазмидой, экспрессирующей антигеномную (т.е. положительной полярности, (+-sense)) РНК VSV, в которой ген гликопротеина G заменен на ген-репортер (pVSVΔG)
и четырьмя добавочными плазмидами, кодирующими белки N (pN.exp), Р (рР.ехр), G (pG.exp), и L (pL.exp), каждый находится под контролем промотора Т7 (Фиг. 1, I).
Фракция экспрессируемой Т7 антигеномной РНК инкапсулируется белком N VSV, и эта инкапсулированная РНК будет служить матрицей для синтеза геномной РНК полимеразным комплексом VSV, состоящим из белков L и Р VSV. Как только геномная РНК произведена, что может произойти только благодаря активности полимеразного комплекса VSV, взаимодействующего с инкапсулированным антигеномом, синтезируются вирусные мРНК, и цикл репликации завершается. В дополнение к большему количеству белков N, Р и L из кодируемого вирусом гена М продуцируется матричный белок (М) VSV. Белок М необходим для конденсации рибонуклеопротеинов и отпочковывания вируса. Поскольку rVSV-ΔG не кодирует вирусный гликопротеин (G), он должен быть представлен in trans для получения инфекционных G-псевдотипированных вирионов rVSV-ΔG. Протокол выделения и титрования G-псевдотипированного rVSV-ΔG (G*ΔG-VSV) разделен на два этапа.
Сначала клетки инфицируют рекомбинантным вирусом осповакцины, экспрессирующим РНК-полимеразу Т7 (vvT7), а затем инфицированные клетки трансфицируют плазмидами N, Р, G, L и pVSV-ΔG в соотношении 3:5:8:1:5. Культуральную среду, кондиционированную трансфицированными клетками и содержащую вирусные частицы, используют для трансфекции другой порции клеток, предварительно трансфицированных плазмидой pCAGGS-G, которая экспрессирует белок G VSV под контролем сильного промотора pol-II. При культивировании этих клеток происходит амплификация вируса, кондиционированная среда через 24-48 часов содержит вирусные частицы rVSV-ΔG
В результате экспрессии трансфицированных в клетку плазмид образуется VSV-ΔG-G, но низкого титра, и, далее, с целью повышения трансдуцирующего титра, полученный вирус добавляют клеткам, предварительно трансфицированным плазмидой, экспрессирующей поверхностный гликопротеин G под контролем сильного промотора (Фиг. 1, II). Затем полученные вирусные частицы очищают и определяют трансдуцирующий титр методом бляшкообразования в клеточном монослое, экспрессирующем гликопротеин G, поскольку образование бляшек возможно только при многоцикловой репликации вируса (Фиг. 1, III-IV). При заражении полученным VSV-ΔG клеток, экспрессирующих на мембране поверхностные гликопротеины (или комплекс гликопротеинов) гетерологичного вируса (G*), образуются псевдовирусы VSV-ΔG-G*. Инфекционность таких частиц - псевдовирусов определяется уже поверхностными белками гетерологичного вируса, и ограничивается лишь одним циклом инфекции, что обеспечивает биологическую безопасность этой системы. Однако для осуществления этого способа требуется использование живого не аттенюированного (не ослабленного) вируса осповакцины, для безопасной работы с которым требуются дополнительные меры по организации работы персонала, хранению вирусных банков, организации помещений. Кроме того, на этапе масштабирования потребуется проведение процедуры, доказывающей, что в конечном лекарственном препарате не содержится остатков вируса осповакцины (как одного из компонентов технологического процесса, для чего потребуется проведение валидации процесса вирусной очистки. Недостатком этого способа также является ограниченное применение получаемого репликативно-неактивного VSV, который может быть использован либо как вектор для доставки трансгена по аналогии с векторами для генной терапии, либо как псевдотипированный вирус, используемый в качестве вакцины, но не как онколитик.
Известен способ получения рекомбинантных вирусных частиц VSV, заключающийся в том, что смесь 1:1 клеток 293T:VeroE6 трансфицируют смесью плазмид, кодирующих pVSVXN2ΔG/EBOVGP, Т7 полимеразу, N, L, Р в соотношениях 8:8:2:1:5, 8:10:2:1:5, 4:4:1:1,2:2,5, 4:4:1:1,2:2,5 и 8:8:2:1:5, в присутствии липофектамина, затем кондиционированную этими клетками среду добавляют к свежей культуре клеток VeroE6 [Wong G, Qiu X. Designing Efficacious Vesicular Stomatitis Virus-Vectored Vaccines Against Ebola Virus. Methods Mol Biol. 2016;1403:245-57. doi: 10.1007/978-1-4939-3387-7_12]. Данный способ предназначен для создания псевдотипированного репликативно активного вируса, который используется в качестве вакцины от вируса Эбола. Первый этап сборки VSV предполагает использование клеток HEK293, которые хорошо трансфицируются, и одновременно с этим в той же культуре клеток Vero, роль которых заключается в репликации VSV. Сам метод плохо стандартизуется, т.к. невозможно рассчитать точное количество клеток в каждой из культур на первом этапе сборки. К тому же на этапе трансфекции используется соотношение, при котором основную генетическую нагрузку на клетки, наравне с коровым вектором с вирусным геномом, несет плазмида, кодирующая Т7 полимеразу, что может негативно сказаться на эффективности первичной сборки VSV, т.к. количество хелперных плазмид добавляется к клеткам в несколько раз меньше. Для наработки (амплификации) используется клеточная культура Vero, которая является адгезионной, а для масштабирования биопроцесса необходима ее адаптация к суспензионному росту. При этом клетки HEK293 и BHK21 легко могут быть переведены в суспензионный формат и чаще других клеточных культур используются для наработки и масштабирования технологии получения рекомбинантных белков и вирусов.
Известен способ, являющийся модификацией предыдущего способа, заключающийся в том, что 1,0×106 клеток HEK293 высевают в лунку 6-луночного планшета за 1 день до трансфекции. На следующий день 5 мкг плазмиды, экспрессирующей вирусный геном с положительной цепью, трансфицируют в клетки HEK293 с использованием TransReagent IT-293 (Mirus Bio) вместе с соответствующим количеством плазмид, экспрессирующих вирусные белки и РНК-полимеразу Т7 (1,5 мкг pCAG-VSVN, 2,5 мкг pCAG-VSVP, 0,5 мкг pCAG-VSVL, 4 мкг pCAG-VSVG и 5 мкг РНК-полимеразы pCAG-T7), и клетки культивируют. Через два дня после трансфекции культуральную жидкость, содержащую некомпетентные к репликации частицы VSV, собирают и инокулируют в клетки BHK-21, трансфицированные pCAG-VSVG, для амплификации вирусных частиц. Культуральную жидкость собирают через 2-3 дня после инокуляции и хранят при -80°С. Титр исходного вируса определяют путем подсчета количества клеток BHK-21 или HEK293, экспрессирующих GFP, после инфицирования [Takahashi K, Yokobayashi Y. Reversible Gene Regulation in Mammalian Cells Using Riboswitch-Engineered Vesicular Stomatitis Virus Vector. ACS Synth Biol. 2019 Sep 20;8(9): 1976-1982]. Данный способ предполагает получение репликативно-неактивного вируса, который не может быть использован в качестве онколитика. К тому же способ предполагает использование генома вируса, где первый ген со стороны 3'-конца генома VSV - это GFP, что может сказаться на эффективности сборки и репликации, т.к. изменение последовательности генов в геноме VSV - это один из способов аттенюации вируса (поэтому этот способ больше подходит для создания вакцин или изучения биологии VSV [Clarke DK, Nasar F, Lee M, Johnson JE, Wright K, Calderon P, Guo M, Natuk R, Cooper D, Hendry RM, Udem SA. Synergistic attenuation of vesicular stomatitis virus by combination of specific G gene truncations and N gene translocations. J Virol. 2007 Feb;81(4):2056-64. doi: 10.1128/JVI.01911-06].
Для создания воспроизводимой, эффективной и безопасной технологии получения рекомбинантных VSV для использования в качестве онколитических вирусов целесообразно не только использовать репликативно-активную форму VSV, что в том числе убирает необходимость предварительной трансфекции культуры клеток для наработки VSV вектором с G-геном, но и подобрать условия проведения трансфекции клеточной культуры для первичной сборки VSV, в том числе соотношение плазмид добавляемых в лунку 6- или 12-луночного планшета (соотношение плазмид по весу), подобрать оптимальное количество плазмидной ДНК, добавляемой клеткам, подобрать клетки для первичной сборки, уйти от необходимости использования хелперного живого вируса для осуществления первого этапа сборки, подобрать количество и/или оптимизировать структуру генетических хелперных конструкций, а также снизить по возможности количество одновременно используемых конструкций или их количество, используемое на этапе трансфекции для уменьшения генетической нагрузки на клетки. Важной частью технологии является потенциальная возможность масштабирования технологического процесса и использование бессывороточных сред для культивирования и очистки лекарственных продуктов на основе VSV.
Технической задачей, на решение которой направлено настоящее изобретение, является решение как минимум одной из вышеуказанных в уровне техники проблем.
Сущность изобретения
Технической решением является использование описанного признаками в пунктах формулы изобретения.
Одной из возможных технических задач, на решение которой направлено настоящее изобретение, являлось создание способа получения рекомбинантного вируса везикулярного стоматита (rVSV) без использования дополнительных живых вирусов (оспа, вирус герпеса или других) для упрощения производственного процесса, в т.ч. за счет снятия ограничений, связанных с необходимостью работы с живым вирусом на первом этапе получения содержащей вирус среды. Другой возможной задачей, на решение которой направлено настоящее изобретение, было создание способа получения рекомбинантного вируса везикулярного стоматита, позволяющего получить достаточное количество вирусных частиц rVSV, не уступающего продуктивности при получении вируса дикого типа.
Задача может решаться тем, что предлагается способ получения рекомбинантного вируса везикулярного стоматита, состоящий из этапа трансфекции и трансдукции. Заявляемый способ характеризуется тем, что на этапе трансфекции клетки, стабильно экспрессирующие ДНК-зависимую РНК-полимеразу бактериофага Т7, трансфицируют с использованием смеси плазмидных экспрессионных векторов и полиэтиленимина в весовом соотношении ДНК: полиэтиленимин 5:1, культивируют, собирают культуральную жидкость. Упомянутая смесь плазмидных экспрессионных векторов содержит четыре плазмидных экспрессионных вектора pCAG-VSVN, pCAG-VSVP, pCAG-VSVL и pCAG-VSVG, каждый из которых кодирует один из белков вируса везикулярного стоматита N, Р, L, G соответственно под контролем промотора CAG, и плазмидный экспрессионный вектор, кодирующий антигеномно-смысловую РНК генома вируса везикулярного стоматита под контролем промотора Т7. Упомянутые векторы, кодирующие белки N, Р, L, G и антигеномно-смысловую РНК генома вируса везикулярного стоматита берут в соотношении 3:5:1:4:8 или 3:5:1:8:10. В одном из вариантов осуществления изобретения, при получении вируса везикулярного стоматита в объеме лунок 12-луночного планшета общее количество плазмидной ДНК при расчете на лунку составляет 10,5 мкг при соотношении векторов, кодирующих белки N, Р, L, G и антигеномно-смысловую РНК генома вируса везикулярного стоматита 3:5:1:4:8. В другом варианте осуществления изобретения, при получении вируса везикулярного стоматита в объеме лунок 12-луночного планшета общее количество плазмидной ДНК при расчете на лунку составляет 6,75 мкг при соотношении векторов, кодирующих белки N, Р, L, G и антигеномно-смысловую РНК генома вируса везикулярного стоматита 3:5:1:8:10.
На этапе трансдукции предварительно выращенные клетки трансдуцируют (заражают) кондиционированной культуральной жидкостью, полученной после культивирования трансфицированных клеток, культивируют трансдуцированные клетки и собирают кондиционированную культуральную жидкость.
В других вариантах осуществления изобретения этап трансдукции повторяют один или более раз с использованием свежих клеток.
В одном из вариантов осуществления изобретения клетки, стабильно экспрессирующие ДНК-зависимую РНК-полимеразу бактериофага Т7, представляют собой клетки линии HEK293TN, стабильно экспрессирующие ДНК-зависимую РНК-полимеразу бактериофага Т7.
В одном из вариантов осуществления изобретения клетки линии HEK293TN, стабильно экспрессирующие ДНК-зависимую РНК-полимеразу бактериофага Т7, получены путем трансдукции клеток линии HEK293TN ретровирусом, несущим ген ДНК-зависимой РНК-полимеразы бактериофага Т7.
В другом варианте осуществления изобретения используют клетки линии BHK21 Т7, стабильно экспрессирующие ДНК-зависимую РНК-полимеразу бактериофага Т7. Одним из возможных способов получения клеток линии BHK21 Т7, стабильно экспрессирующих ДНК-зависимую РНК-полимеразу бактериофага Т7, может быть трансдукция клеток линии BHK21 ретровирусом, несущим ген ДНК-зависимой РНК-полимеразы бактериофага Т7.
В одном из вариантов осуществления изобретения смесь плазмидных экспрессионных векторов дополнительно содержит плазмидный экспрессионный вектор, кодирующий ДНК-зависимую РНК-полимеразу бактериофага Т7.
В одном из вариантов осуществления изобретения смесь плазмидных экспрессионных векторов содержит плазмидный экспрессионный вектор, кодирующий ДНК-зависимую РНК-полимеразу бактериофага Т7, при этом векторы, кодирующие белки вируса везикулярного стоматита N, Р, L, G, вектор, кодирующий ДНК-зависимую РНК-полимеразу бактериофага Т7, и вектор, кодирующий антигеномно-смысловую РНК генома вируса везикулярного стоматита, содержатся в упомянутой смеси в соотношении 3:5:1:4:5:8 или 3:5:1:8:10:10.
В одном из вариантов осуществления изобретения плазмидный экспрессионный вектор, кодирующий антигеномно-смысловую РНК генома вируса везикулярного стоматита, содержит гетерологичный ген (трансген), кодирующий белок интереса.
В одном из вариантов осуществления изобретения белок интереса представляет собой зеленый флюоресцентный белок, или химерный белок.
В одном из вариантов осуществления изобретения плазмидный экспрессионный вектор, кодирующий белок вируса везикулярного стоматита N, представляет собой вектор, содержащий в соответствии с физической и генетической картой, представленной на Фиг. 6В, следующие элементы:
энхансер цитомегаловирусного промотора CMV enhancer;
промотор бета-актина цыпленка Chicken β-actin promoter;
химерный интрон, увеличивающий экспрессию трансгена Chimeric intron;
последовательность гена белка VSV-N нуклеокапсида вируса везикулярного стоматита VSV-N;
сигнал полиаденилирования β-globin poly(A) signal;
Lac-оператор Lac operator;
Lac-промотор Lac promoter;
промотор SV40 SV40 promoter;
участок начала репликации SV40 ori;
сигнал полиаденилирования SV40 Poly(A)signal;
участок начала репликации ori;
ген устойчивости к ампициллину AmpR;
промотор гена устойчивости к ампициллину AmpR promoter.
В одном из вариантов осуществления изобретения плазмидный экспрессионный вектор, кодирующий белок вируса везикулярного стоматита N, содержит последовательность SEQ Id No.: 5.
В одном из вариантов осуществления изобретения плазмидный экспрессионный вектор, кодирующий белок вируса везикулярного стоматита Р, представляет собой вектор, содержащий в соответствии с физической и генетической картой, представленной на Фиг. 6Б, следующие элементы:
энхансер цитомегаловирусного промотора CMV enhancer;
промотор бета-актина цыпленка Chicken β-actin promoter;
химерный интрон, увеличивающий экспрессию трансгена Chimeric intron;
последовательность гена белка VSV-P фосфопротеина вируса везикулярного стоматита VSV-P;
сигнал полиаденилирования β-globin poly(A) signal;
Lac-оператор Lac operator;
Lac-промотор Lac promoter;
промотор SV40 SV40 promoter;
участок начала репликации;
сигнал полиаденилирования SV40 Poly(A)signal;
участок начала репликации ori;
ген устойчивости к ампициллину AmpR;
промотор гена устойчивости к ампициллину AmpR promoter.
В одном из вариантов осуществления изобретения плазмидный экспрессионный вектор, кодирующий белок вируса везикулярного стоматита Р, содержит последовательность SEQ Id No.: 6.
В одном из вариантов осуществления изобретения плазмидный экспрессионный вектор, кодирующий белок вируса везикулярного стоматита L, представляет собой вектор, содержащий в соответствии с физической и генетической картой, представленной на Фиг. 6А, следующие элементы:
энхансер цитомегаловирусного промотора CMV enhancer;
промотор бета-актина цыпленка Chicken β-actin promoter;
химерный интрон, увеличивающий экспрессию трансгена Chimeric intron;
последовательность гена белка VSV-L большой субъединицы полимеразы вируса везикулярного стоматита VSV-L;
сигнал полиаденилирования β-globin poly(A) signal;
Lac-оператор Lac operator;
Lac-промотор Lac promoter;
промотор SV40 SV40 promoter;
участок начала репликации SV40 ori;
сигнал полиаденилирования SV40 Poly(A)signal;
участок начала репликации ori;
ген устойчивости к ампициллину AmpR;
промотор гена устойчивости к ампициллину AmpR promoter.
В одном из вариантов осуществления изобретения плазмидный экспрессионный вектор, кодирующий белок вируса везикулярного стоматита Р, содержит последовательность SEQ Id No.: 4.
Фигуры
Изобретение иллюстрируются следующими графическими материалами:
На Фиг. 1 представлена схема получения rVSV-ΔG-G и его амплификация. (А) Схема кДНК, кодирующей антигеном rVSV-ΔG-GFP (pVSV-ΔG-GFP или pVSV-ΔG*). Консервативные регуляторные последовательности транскрипции VSV (октагон = стоп-последовательность; An = последовательность полиаденилирования; стрелка = промотор транскрипции или стартовая последовательность) показаны над диаграммой. Под диаграммой указаны последовательности промотора (Т7) и терминатора РНК-полимеразы бактериофага Т7 (Тφ) и рибозима вируса гепатита дельта (δ-RBZ) с сайтом разрезания. (В) I. Получение VSV-ΔG-G из плазмид. Первоначально клетки котрансфицируют плазмидой pVSV-ΔG-GFP и четырьмя вспомогательными (хелперными) плазмидами, кодирующими белки VSV N, Р, G и L. II-IV. Амплификация rVSV-ΔG-GFP и определение трансдуцирующего титра сборки, роста и анализа rVSV-ΔG. Рисунок цитирован в работе Whitt MA. Generation of VSV pseudotypes using recombinant ΔG-VSV for studies on virus entry, identification of entry inhibitors, and immune responses to vaccines // Journal of Virological Methods. - 2010. - T. 169 - №. 2. C. 365-374, для которой был предоставлен CS Robison [Robison CS. Microbiology & Immunology, Vol. Ph.D. Memphis: University of Tennessee Health Science Center; 2001. Assembly and fusion-potentiation domains of the vesicular stomatitis virus glycoprotein and their utility in the development of cell-type-targeted rhabdoviral vectors; p. 157].
На Фиг 2 представлена микрофотография монослоя клеток HEK293TN, трансфицированных плазмидой pEGFP после 48 часов после трансфекции.
На Фиг. 3 представлен анализ экспрессии вирусных генов после трансфекции методом RT-PCR. А - трансфекция с использованием исходной pVSV-dG-GFP коровой плазмиды для наработки рекомбинантных VSV (rVSV) в HEK293 клетках; Б трансфекция с использованием измененной нами коровой плазмиды pVSV-dG-GFP для наработки рекомбинантных VSV (rVSV) в HEK293 клетках; В - трансфекция с использованием измененной нами коровой плазмиды pVSV-dG-GFP для наработки рекомбинантных VSV (rVSV) в Vero76 клетках.
На Фиг. 4 представлена микрофотография клеток HEK293TN после трансфекции pCAGGS-G. Контуром отмечены области слияния клеток, вызванного экспрессией G-гена VSV.
На Фиг. 5 представлена структурная карта плазмидного корового вектора pVSV-G-GFP-dM51.
На Фиг. 6 представлены структурные карты хелперных плазмид для сборки rVSV. А - pCAG-VSVL, Б - pCAG-VSVP, В - pCAG-VSVN.
Ha Фиг. 7 демонстрируются микрофотографии культуры клеток HEK293TN через 48 часов после трансфекции (pCAG-N/P/L + pVSV-G-GFP-d51 + T7). Справа - положительный контроль трансфекции.
На Фиг. 8 представлены результаты анализа продуктов ПЦР-реакции в процессе скрининга колоний клеток HEK293TN, стабильно экспрессирующих Т7-РНК-полимеразу. Маркер молекулярного веса на правой дорожке.
На Фиг. 9 представлены результаты анализа интенсивности люминесценции после трансфекции клеток HEK293TN-T7. Mock - трансфекция плазмидой pEGFPN3; Luc - трансфекция плазмидой pS_5-Mod-LucF-3-Mod; T7Luc трансфекция плазмидами pCAG-T7pol+pS_5-Mod-LucF-3-Mod.
На Фиг. 10 представлены микрофотографии клеток HEK293TN-T7 после трансфекции в оптимизированных условиях. Слева направо: трансфекция для наработки rVSV, положительный контроль, отрицательный контроль.
На Фиг. 11 представлена визуализация цитопатического эффекта rVSV и конечный титр вируса в культуральной жидкости после культивирования в культуральных флаконах площадью 150 см2.
На Фиг. 12 представлены микрофотографии клеток BHK21 трансдуцированных rVSV в предельном разведении в 107 раз в лунках при наличии ЦПЭ (слева) и без ЦПЭ (справа).
На Фиг. 13 демонстрируется подтверждение подлинности rVSV с помощью ПЦР, в ходе которой амплифицируется встроенный в геном rVSV участок ДНК с геном GFP (А), и электрофореза в ПААГ (Б).
На Фиг. 14 демонстрируется участок карты плазмидной ДНК pVSV-G-GFP-d51, на которой отмечены места отжига универсальных праймеров (vsvG_end_f и vsvL_start-r), которые используются для анализа наличия дополнительных генов между генами G и L (на изображенном участке плазмиды в указанную область встроен ген, кодирующий GFP).
На Фиг. 15 демонстрируется результат анализа ПЦР-амплификации фрагмента, фланкированного универсальными праймерами vsvG_end_f и vsvL_start-r, который не содержит ген GFP (фрагмент ~450 п.н.).
На Фиг. 16 представлена структурная карта коровой плазмиды, содержащей последовательность SEQ Id No: 7.
На Фиг. 17 представлена структурная карта коровой плазмиды, содержащей последовательность SEQ Id No: 8.
Детальное описание изобретения
Если не указано иначе, предполагается, что все термины, обозначения и другие научные термины, используемые в данной заявке, имеют значения, которые обычно понимают специалисты в области, к которой относится настоящее изобретение. В некоторых случаях определения терминов с общепринятыми значениями приведены в данной заявке для ясности и/или для быстрой справки и понимания, и включение таких определений в настоящее описание не должно истолковываться как наличие существенного отличия значения термина от обычно подразумеваемого в данной области.
Кроме того, если по контексту не требуется иное, термины в единственном числе включают в себя термины во множественном числе, и термины во множественном числе включают в себя термины в единственном числе. Как правило, используемая классификация и методы культивирования клеток, молекулярной биологии, иммунологии, микробиологии, генетики, аналитической химии, химии органического синтеза, медицинской и фармацевтической химии, а также гибридизации и химии белка и нуклеиновых кислот, описанные в настоящем документе, хорошо известны специалистам и широко применяются в данной области. Ферментативные реакции и способы очистки осуществляют в соответствии с инструкциями производителя, как это обычно осуществляется в данной области, или как описано в настоящем документе.
Одной из возможных технических задач, на решение которой направлено настоящее изобретение, являлось создание способа получения рекомбинантного вируса везикулярного стоматита (rVSV) без использования дополнительных живых вирусов (оспа, вирус герпеса или других) для упрощения производственного процесса, в т.ч. за счет снятия ограничений, связанных с необходимостью работы с живым вирусом на первом этапе получения содержащей вирус среды. Другой возможной задачей, на решение которой направлено настоящее изобретение, было создание способа получения рекомбинантного вируса везикулярного стоматита, позволяющего получить достаточное количество вирусных частиц rVSV, не уступающего продуктивности при получении вируса дикого типа.
Для создания технологии получения rVSV необходимо было проанализировать возможные пути оптимизации классического протокола: ре-дизайн плазмид и уменьшение общего количества плазмид (например, за счет pCAGGS-G) для снижения нагрузки ДНК на клетки при трансфекции.
Термин «рекомбинантный вирус везикулярного стоматита» или «рекомбинантный везикуловирус», далее обозначаемый также как «rVSV», обозначает вирус везикулярного стоматита, который получен методами генной инженерии и необязательно содержит трансген, кодирующий белок интереса.
В одном из вариантов осуществления настоящего изобретения предлагается способ получения рекомбинантного вируса везикулярного стоматита, состоящий из этапа трансфекции и трансдукции. Термин «трансфекция» обозначает процесс введения в эукариотические клетки чужеродного (трансгенного) генетического материала невирусным способом. Чаще всего для трансфекции используют плазмидные экспрессионные векторы, или плазмиды, несущие целевой гетерологичный ген (трансген) и регуляторные элементы, необходимые для его экспрессии. Термин «трансдукция» обозначает процесс переноса генетического материала между клетками при помощи вирусов или вирусных векторов. Трансдукция - это рутинный инструмент, используемый молекулярными биологами для стабильного введения чужеродного гена в геном клетки-хозяина (как бактерии, так и клетки млекопитающих). Поскольку в процессе трансдукции используются вирусы, этот процесс также называют «заражением» или «инфицированием».
Первый этап сборки рекомбинантного вируса rVSV-G-GFP-dM51 включал трансфекцию клеток полученной ранее линии HEK293-Т7 четырьмя плазмидами pCAG-VSVN, pCAG-VSVP, pCAG-VSVG и pCAG-VSVL, каждая из которых содержит кДНК, кодирующую один из белков VSV N, VSV Р, VSV G, VSV L соответственно, и коровой плазмидой pVSV-G-GFP-dM51, экспрессирующей антигеномно-смысловую (или положительно-смысловую) РНК генома VSV под контролем промотора Т7. Трансфекцию проводили с добавлением либо без добавления плазмиды, содержащей ДНК-зависимую РНК-полимеразу бактериофага Т7. На исследовательской стадии для облегчения детекции эффективности способа в качестве гена интереса использовали ген, кодирующий зеленый флуоресцентный белок (GFP). Гены всех белков в вспомогательных плазмидах находятся под контролем CAG промотора в отличие от плазмид, использованных в известном протоколе [Whitt MA. Generation of VSV pseudotypes using recombinant ΔG-VSV for studies on virus entry, identification of entry inhibitors, and immune responses to vaccines // Journal of Virological Methods. - 2010. - T. 169 - №. 2. C. 365-374], в котором используется T7 промотор.
Заявляемый способ характеризуется тем, что на этапе трансфекции клетки, стабильно экспрессирующие ДНК-зависимую РНК-полимеразу бактериофага Т7, трансфицируют с использованием смеси плазмидных экспрессионных векторов и полиэтиленимина в весовом соотношении ДНК: полиэтиленимин 5:1, культивируют, собирают кондиционированную культуральную жидкость. Термины «кондиционированная культуральная жидкость» или «кондиционированная среда» обозначают культуральную среду после культивирования в ней клеток, содержащую компоненты, секретируемые клетками, в частности, в отношении настоящего изобретения кондиционированная культуральная жидкость содержит вирусные частицы рекомбинантного вируса везикулярного стоматита.
Термины «питательная среда для культивирования клеток», «среда для культивирования клеток», «культуральная среда» или коротко «среда» обозначают многокомпонентные жидкости или плотные гели, созданные специально для выращивания клеток разного типа. В их состав входят различные аминокислоты, соли, витамины, гормоны, факторы прикрепления и роста, смешанные в определенных пропорциях. В настоящее время в уровне техники известны и доступны как рецепты культуральных сред, так и коммерческие готовые культуральные среды. Выбор конкретной культуральной среды определяется задачами, для которых она будет в дальнейшем использована, доступностью, ценой и опытом конкретной лаборатории и не ограничивает объем настоящего изобретения.
Упомянутая смесь плазмидных экспрессионных векторов содержит четыре плазмидных экспрессионных вектора pCAG-VSVN, pCAG-VSVP, pCAG-VSVL и pCAG-VSVG, каждый из которых кодирует один из белков вируса везикулярного стоматита N, Р, L, G соответственно под контролем промотора CAG, и плазмидный экспрессионный вектор, кодирующий антигеномно-смысловую РНК генома вируса везикулярного стоматита под контролем промотора CAG. Упомянутые векторы, кодирующие белки N, Р, L, G и антигеномно-смысловую РНК генома вируса везикулярного стоматита берут в соотношении 3:5:1:4:8 или 3:5:1:8:10. Термин «плазмидные экспрессионные векторы» или «плазмиды» обозначает небольшие молекулы ДНК, физически обособленные от хромосом и способные к автономной репликации, содержащие чужеродный генетический материал и регуляторные элементы, необходимые для его экспрессии.
В одном из вариантов осуществления изобретения используют хелперные плазмидные векторы или хелперные плазмиды. Термин «вспомогательная плазмида» или «хелперная плазмида» обозначает плазмидный экспрессионный вектор, несущий гены, экспрессия которых необходима для сборки вирусных частиц. В частности, хелперными плазмидами являются плазмиды pCAG-VSVN, pCAG-VSVP, pCAG-VSVL и pCAG-VSVG, несущие гены VSV, кодирующие структурные вирусные белки.
В качестве вспомогательных плазмид могут использоваться любые известные из уровня техники плазмиды, в том числе коммерчески доступные плазмидные экспрессионные векторы, предназначенные для сборки вирусных частиц вируса везикулярного стоматита. Вспомогательные плазмиды известны специалисту в этой области техники, и могут быть как собраны de novo, так и приобретены готовыми, в том числе в составе готовых комплектов [см. для примера https://www.kerafast.com/item/583/set-of-helper-plasmids-vsv-n-vsv-p-vsv-1-vsv-g, дата обращения 26.12.2022].В одном из вариантов осуществления изобретения плазмидный экспрессионный вектор, кодирующий белок вируса везикулярного стоматита N, представляет собой вектор, содержащий в соответствии с физической и генетической картой, представленной на Фиг. 6В, следующие элементы:
энхансер цитомегаловирусного промотора CMV enhancer;
промотор бета-актина цыпленка Chicken β-actin promoter;
химерный интрон, увеличивающий экспрессию трансгена Chimeric intron;
последовательность гена белка VSV-N нуклеокапсида вируса везикулярного стоматита VSV-N;
сигнал полиаденилирования β-globin poly(A) signal;
Lac-оператор Lac operator;
Lac-промотор Lac promoter;
промотор SV40 SV40 promoter;
участок начала репликации SV40 ori;
сигнал полиаденилирования SV40 Poly(A)signal;
участок начала репликации ori;
ген устойчивости к ампициллину AmpR;
промотор гена устойчивости к ампициллину AmpR promoter.
В одном из вариантов осуществления изобретения плазмидный экспрессионный вектор, кодирующий белок вируса везикулярного стоматита N, содержит последовательность SEQ Id No.: 5.
В одном из вариантов осуществления изобретения плазмидный экспрессионный вектор, кодирующий белок вируса везикулярного стоматита Р, представляет собой вектор, содержащий в соответствии с физической и генетической картой, представленной на Фиг. 6Б, следующие элементы:
энхансер цитомегаловирусного промотора CMV enhancer;
промотор бета-актина цыпленка Chicken β-actin promoter;
химерный интрон, увеличивающий экспрессию трансгена Chimeric intron;
последовательность гена белка VSV-P фосфопротеина вируса везикулярного стоматита VSV-P;
сигнал полиаденилирования β-globin poly(A) signal;
Lac-оператор Lac operator;
Lac-промотор Lac promoter;
промотор SV40 SV40 promoter;
участок начала репликации SV40 ori;
сигнал полиаденилирования SV40 Poly(A)signal;
участок начала репликации ori;
ген устойчивости к ампициллину AmpR;
промотор гена устойчивости к ампициллину AmpR promoter.
В одном из вариантов осуществления изобретения плазмидный экспрессионный вектор, кодирующий белок вируса везикулярного стоматита Р, содержит последовательность SEQ Id No.: 6.
В одном из вариантов осуществления изобретения плазмидный экспрессионный вектор, кодирующий белок вируса везикулярного стоматита L, представляет собой вектор, содержащий в соответствии с физической и генетической картой, представленной на Фиг. 6А, следующие элементы:
энхансер цитомегаловирусного промотора CMV enhancer;
промотор бета-актина цыпленка Chicken β-actin promoter;
химерный интрон, увеличивающий экспрессию трансгена Chimeric intron;
последовательность гена белка VSV-L большой субъединицы полимеразы вируса везикулярного стоматита VSV-L;
сигнал полиаденилирования β-globin poly(A) signal;
Lac-оператор Lac operator;
Lac-промотор Lac promoter;
промотор SV40 SV40 promoter;
участок начала репликации SV40 ori;
сигнал полиаденилирования SV40 Poly(A)signal;
участок начала репликации ori;
ген устойчивости к ампициллину AmpR;
промотор гена устойчивости к ампициллину AmpR promoter.
В одном из вариантов осуществления изобретения плазмидный экспрессионный вектор, кодирующий белок вируса везикулярного стоматита Р, содержит последовательность SEQ Id No.: 4.
Клетки, стабильно экспрессирующие ДНК-зависимую РНК-полимеразу бактериофага Т7, также сокращенно называемую в настоящем описании «Т7 полимераза», могут быть получены известными специалисту в данной области способами, в том числе можно использовать коммерчески доступные клетки, например, клетки линии BHK21, HEK293, А549, HeLa, Vero и др. Выбор конкретной клеточной линии определяется задачами, для которых она будет в дальнейшем использована, доступностью, ценой и опытом конкретной лаборатории и не ограничивает объем настоящего изобретения.
В одном из вариантов осуществления изобретения используют клетки линии HEK293TN, стабильно экспрессирующие ДНК-зависимую РНК-полимеразу бактериофага Т7. Одним из возможных способов получения клеток линии HEK293TN, стабильно экспрессирующих ДНК-зависимую РНК-полимеразу бактериофага Т7, может быть трансдукция клеток линии HEK293TN ретровирусом, несущим ген ДНК-зависимой РНК-полимеразы бактериофага Т7.
В другом варианте осуществления изобретения используют клетки линии BHK21 Т7, стабильно экспрессирующие ДНК-зависимую РНК-полимеразу бактериофага Т7. Одним из возможных способов получения клеток линии BHK21 Т7, стабильно экспрессирующих ДНК-зависимую РНК-полимеразу бактериофага Т7, может быть трансдукция клеток линии BHK21 ретровирусом, несущим ген ДНК-зависимой РНК-полимеразы бактериофага Т7.
В одном из вариантов осуществления изобретения смесь плазмидных экспрессионных векторов дополнительно содержит плазмидный экспрессионный вектор, кодирующий ДНК-зависимую РНК-полимеразу бактериофага Т7, при этом векторы, кодирующие белки вируса везикулярного стоматита N, Р, L, G, вектор, кодирующий ДНК-зависимую РНК-полимеразу бактериофага Т7, и вектор, кодирующий антигеномно-смысловую РНК генома вируса везикулярного стоматита, содержатся в упомянутой смеси в соотношении 3:5:1:4:5:8 или 3:5:1:8:10:10. Добавление вектора, кодирующего Т7 полимеразу, приводит к незначительному увеличению эффективности экспрессии генов с плазмид в ходе сборки вирусных части на этапе трансфекции (данные получены по сигналу GFP), однако мало влияет на результат последующей амплификации вируса на этапе трансдукции.
Трансфекция плазмидами в способе по настоящему изобретению в указанном соотношении позволила стабильно получать от 50-70% трансфицированных клеток, что позволяет использовать кондиционированную среду от этих клеток для следующего этапа трансдукции. Более низкая эффективность трансфекции приводит к необходимости более длительного (3 и более суток) культивирования адгезионных клеток после трансдукции на этапе амплификации, что приводит к достижению клетками полной конфлюэнтности без выраженного цитопатического эффекта, падению их жизнеспособности и снижению (или отсутствию) конечного титра rVSV. Альтернативой увеличению длительности культивирования может быть непредсказуемое увеличение количества пассажей, что увеличивает общую длительность этапа наработки и материальные затраты на процесс.
На этапе трансдукции свежие клетки, например, клетки линии BHK21, или HEK293, Vero, HeLa, А459, заражают (трансдуцируют) кондиционированной культуральной жидкостью, полученной после культивирования трансфицированных клеток, культивируют трансдуцированные клетки и собирают кондиционированную культуральную жидкость. Выбор конкретной клеточной линии определяется задачами, для которых она будет в дальнейшем использована, доступностью, ценой и опытом конкретной лаборатории и не ограничивает объем настоящего изобретения. В результате трансдукции клетки заражаются вирусными частицами, что приводит к размножению и амплификации вирусных частиц. Частицы выделяются в культуральную жидкость, которую можно впоследствии использовать как для дополнительной трансдукции новой порции клеток, так и для выделения и очистки вирусных частиц для использования в терапевтических целях, либо изучения активности вирусных частиц.
В других вариантах осуществления изобретения этап трансдукции повторяют один или более раз с использованием новой порции клеток. Дополнительные этапы трансдукции можно повторять необходимое количество раз в зависимости от цели наработки, это приводит к дополнительной амплификации вирусных частиц.
Способ позволяет получать рекомбинантный вирус везикулярного стоматита, что делает возможным получение большого количества вирусных частиц, например, содержащих гетерогенный генетический материал, кодирующий белки интереса.
Одним из белков интереса может быть зеленый флуоресцентный белок (GFP), который может служить в исследовательских целях для визуализации клеток, в которых идет экспрессия генетического материала вируса везикулярного стоматита, для выявления зараженных клеток и т.п. Для усиления онколитических свойств вируса везикулярного стоматита геном вируса может содержать белки, в том числе химерные, зарекомендовавшие себя как эффективные агенты иммунотерапии опухолей. В частности, полученный заявленным способом рекомбинантный вирус везикулярного стоматита, экспрессирующий химерный белок IL12-GMCSF демонстрирует поразительно высокую онколитическую активность по сравнению с аналогичным вирусом, не экспрессирующим упомянутый химерный белок.
Термин «засевание», «пассирование», «высевание» клеток обозначает отбор небольшого количества клеток (от 1/2 до 1/5 от общего количества) для выращивания в другом лабораторном сосуде.
Термин «цитопатическое действие» или «цитопатический эффект» (ЦПЭ) обозначает возникновение дегенеративных изменений в клеточных культурах, обычно связанное с размножением вирусов. В частности, одним из внешних признаков цитопатического эффекта при инфекции VSV является округление клеток, что связано с индукцией апоптоза [Kopecky SA, Lyles DS. The cell-rounding activity of the vesicular stomatitis virus matrix protein is due to the induction of cell death. J Virol. 2003 May;77(9):5524-8. doi: 10.1128/jvi.77.9.5524-5528.2003].
Таким образом, способ по настоящему изобретению позволяет получать вирусные частицы рекомбинантного вируса везикулярного стоматита в титре, сравнимом с инфекционным титром вируса везикулярного стоматита дикого типа. При этом способ по настоящему изобретению не требует использования дополнительных живых вирусов. rVSV, полученный способом по настоящему изобретению, также не отличается от вируса дикого типа по динамике накопления в клетках [Hebert CG, DiNardo N, Evans ZL, et al. Rapid quantification of vesicular stomatitis virus in Vero cells using Laser Force Cytology. Vaccine. 2018 Oct 1;36(41):6061-6069. doi: 10.1016/j.vaccine.2018.09.002].
Сущность и промышленная применимость изобретения поясняются следующими примерами:
Пример 1. Получение rVSV-dG-GFP по стандартному протоколу и проверка работоспособности плазмид.
В качестве исходных векторов для наработки rVSV использовали экспрессионные плазмиды производства Kerafast: pVSV-dG-GFP 2.6 (cat# EH1026), pBS-N-ФТ-Kan (cat# EH1013), pBS-G-OT-Kan (cat# EH1016), pBS-L-ФТ-Amp (cat# EH1015), pBS-P-ФТ-Amp (cat# EH1014), pCAGGS-G-Kan Kerafast (cat# EH1017), а также плазмиду pCAG-T7pol производства Addgene (cat# 59926). Для трансформации бактерий электропорацией и амплификации плазмид использовали бактериальные штаммы Escherichia coli DH5-alpha, NEB® Stable, XL10-Gold®.
Для подтверждения работоспособности плазмид получали репликативно-неактивный rVSV (rVSV-dG-GFP), ориентируясь на условия, представленные в протоколе, на который ссылаются производители плазмид (Kerafast) [Whitt MA. Generation of VSV pseudotypes using recombinant ΔG-VSV for studies on virus entry, identification of entry inhibitors, and immune responses to vaccines // Journal of Virological Methods. - 2010. T. 169 - №. 2. C. 365-374], но с использованием плазмиды, кодирующей Т7 полимеразу, вместо вируса осповакцины.
Для этого по 1,5 мл суспензии клеток HEK293TN (0,5 млн/мл) вносили во все лунки одного 6-луночного планшета из расчета 750 000 клеток на лунку (при использовании 12-луночного планшета во всех экспериментах брали в два раз меньше клеток) за день до трансфекции. Время рассчитывали так, чтобы конфлюэнтность клеток в момент трансфекции на следующий день составила 70-80%. На следующий день за два часа до трансфекции из всех 6 лунок планшета отбирали среду с помощью одноканального дозатора и вносили среду DMEM+2%FBS+2mM L-Gln в объеме 2 мл на лунку. В стерильную пробирку объемом 1,5 мл вносили плазмиды с генами, кодирующими N, Р, G, L и Т7 полимеразу, а также плазмиду pVSV-dG-GFP, кодирующую геномом вируса, в соотношении 3:5:8:1:5:5 мкг каждой плазмиды соответственно. К смеси плазмид добавляли 520 мкл DMEM и 162 мкл полиэтиленимина (PEI, 1 мг/мл в стерильной воде), аккуратно смешивали и инкубировали около 10 минут при комнатной температуре. К клеткам с 2 мл среды DMEM+2%FBS+2mM L-Gln добавляли по каплям трансфицирующую смесь и после культивировали при температуре (37±1)°С в увлажненном воздухе с 5% содержанием СО2. После инкубации в течение ночи провели замену культуральной среды на свежую среду DMEM+2%FBS+2mM L-Gln. Далее кондиционированную культуральную среду собирали, переносили в стерильные центрифужные пробирки объемом 1,5 мл, центрифугировали в течение 15 мин при 400 g, переносили супернатанты в стерильные пробирки и хранили при 4°С или 80°С.
Для амплификации rVSV-dG-GFP (второй этап) проводили трансдукцию клеток полученными на первом этапе вирусными частицами. Для этого содержащую вирусы культуральную жидкость, полученную на первом этапе, добавляли к заранее подготовленным клеткам HEK293TN (трансфицированным плазмидой pCAGGS-G-Kan, как описано в статье Whitt. 2010) в различных разведениях и инкубировали клетки при температуре (37±1)°С в увлажненном воздухе с 5% содержанием СО2. В процессе эксперимента через сутки после трансфекции и через 24 и 48 часов после трансдукции проводили флуоресцентный анализ клеток на инвертированном микроскопе. В результате флуоресцентного анализа ни на этапе после трансфекции, ни на этапах после трансдукции через 24, 48 и даже 120 часов не наблюдали GFP-положительных (GFP+) клеток, несмотря на наличие в трансфекционной смеси плазмиды с Т7 полимеразой, которая должна запускать экспрессию генов, находящихся под контролем Т7 промотора, включая ген, кодирующий GFP в коровом векторе pVSV-dG-GFP 2.6. С помощью проточной цитометрии также не удалось детектировать наличие GFP+HEK293TN клеток. Незначительную флуоресценцию, которую наблюдали в том числе и в контроле, объясняли остаточной флуоресценцией мертвых открепившихся клеток.
При этом в положительном контроле трансфекции с использованием вектора с геном GFP (pEGFP-N3, Addgene, кат. Номер 6080-1) наблюдали высокий уровень флуоресценции (Фиг. 2).
Для выяснения причин отсутствия GFP+ сигнала на этапах трансфекции провели анализ уровня экспрессии трех генов методом РТ-ПЦР, анализ изменения фенотипа клеток после трансфекции плазмидой с G-геном, а также повторили эксперимент с использованием адгезионных культур клеток BHK21, HeLa, Vero, А549. Использование альтернативных клеточных культур не привело к возникновению флуоресцентного сигнала.
Для оценки эффективности транскрипции проводили трансфекцию 2 клеточных линий, HEK293TN и Vero76. Для анализа экспрессии вирусных генов после трансфекции использовали метод ПЦР в реальном времени (RT-PCR, Фиг. 3).
Во всех вариантах наблюдалась экспрессия всех трех проверенных вирусных генов (VSV-L, VSV-M и VSV-P). Нормирование полученных данных проводили по данным экспрессии гена устойчивости к ампициллину (Amp) и гена «домашнего хозяйства», глицеральдегид-3-фосфатдегидрогеназы (GAPDH).
Для проверки работоспособности плазмиды pCAGGS-G проводили трансфекцию линии HEK293-TN в аналогичных условиях. Трансфекция, как и ожидалось, приводит к образованию синцития, что говорит о ее работоспособности (Фиг. 4).
Дополнительно провели полное секвенирование всех плазмид по стандартным протоколам и не выявили отличий от заявленных последовательностей.
Таким образом, для создания технологии получения rVSV необходимо было проанализировать возможные пути оптимизации классического протокола: ре-дизайн плазмид и уменьшение общего количества плазмид (например, за счет pCAGGS-G) для снижения нагрузки ДНК на клетки при трансфекции.
Пример 2. Получение новых хелперных плазмид для проведения первого этапа получения rVSV - трансфекции.
При работе с плазмидной ДНК и для получения новых плазмид использовали стандартные протоколы генетической инженерии, перечисленные далее. Для трансформации и амплификации плазмид использовали бактериальные штаммы Escherichia coli (DH5-alpha, NEB® Stable, XL10-Gold®). Генетическую трансформацию, гидролиз эндонуклеазами рестрикции, лигирование и гель-электрофорез проводили в стандартных условиях [Green, М. and Sambrook, J. (2012) Molecular Cloning: A Laboratory Manual. 4th Edition, Vol. II, Cold Spring Harbor Laboratory Press, New York]. Плазмидную ДНК из бактериальных клеток Е. coli и линеаризованные фрагменты ДНК из агарозных гелей выделяли коммерческими наборами «Plasmid Miniprep», «Cleanup Mini» (ЗАО Евроген, Россия) и «Plasmid mini/midi Kits» (QIAGEN Sciences Inc, США). Плазмиды проверяли с помощью рестрикционного анализа и последующего электрофореза в агарозном геле и методом секвенирования по Сэнгеру. Для внесения мутации в последовательности генов использовали методику инвертированной ПЦР (reverse PCR). Полученный ПЦР-продукт вырезали из геля, обрабатывали Т4 полинуклеотидкиназой и самолигировали фрагмент на самого себя. Полученную лигазную смесь использовали для трансформации клеток Е. coli. Для ПЦР-амплификации фрагментов использовали полимеразу Q5 (NEB). Реакционную смесь готовили согласно рекомендациям производителя. Продукты реакции визуализировали и разделяли электрофоретически в 1% агарозном геле. Полученные ПЦР-фрагменты вырезали из геля, выделили и очистили с помощью набора Cleanup Standard (Евроген). Для анализа колоний-трансформантов проводили ПЦР-скрининг на колониях с использованием праймеров для секвенирования и набора Screen mix (Евроген). Реакционную смесь готовили согласно рекомендациям производителя. Продукты реакции визуализировали и разделяли электрофоретически в 1% агарозном геле. Секвенирование по Сэнгеру осуществляли на амплификаторе ABI 3500 Genetic Analyzer (Applied Biosystems, США) при стандартных условиях и с использованием реагентов, рекомендованных производителем Applied Biosystems. Олигонуклеотидные последовательности проверяли OligoAnalyzer™ Tool [https://www.idtdna.com/pages/tools/oligoanalyzer, дата обращения 15.12.2022]. Длину продукта оценивали с помощью выравнивания на геномную ДНК GRch38/hg38 с помощью Genome Browser [https://genome.ucsc.edu/, дата обращения 15.12.2022].
Первым этапом подготовки плазмид к наработке rVSV было получение методом сайт-направленного мутагенеза корового вектора с геномом VSV, в который введена мутация dM51 в гене М. Коровый вектор - плазмидный экспрессионный вектор, кодирующий антигеномно-смысловую РНК генома вируса везикулярного стоматита под контролем промотора Т7. При попадании VSV в здоровые клетки белок М с делецией в гене М не подавляет экспрессию интерферонов, что делает такую модификацию полезной с точки зрения безопасности терапевтических VSV.
Для внесения мутации в последовательность гена М использовали методику инвертированной ПЦР, используя в качестве матрицы полноразмерную плазмиду pVSV-dG-GFP 2.6 (Kerafast) и праймеры:
прямой: GACACCTATGATCCGAATCAATTAAGATATGAGA
обратный: CTCGTCAACTCCAAAATAGGATTTGTCAATTGGA
Полученный ПЦР-продукт далее вырезали из геля, обрабатывали Т4 полинуклеотид-киназой, и самолигировали фрагмент на самого себя. Полученную лигазную смесь использовали для трансформации клеток Е. coli.
В результате была получена плазмида pVSV-dG-GFP-dM51. Далее по сайтам NheI-XbaI в плазмиду была клонирована последовательность гена G из плазмиды pCAGGS-G и получен базовый коровый вектор, необходимый для получения репликативно-активного rVSV pVSV-G-GFP-dM51 (Фиг. 5).
Далее мы получили 3 вектора, в которых экспрессия генов L, N и Р контролируется CAG-промотором. Для этого в вектор pCAG-T7 клонировали последовательности генов VSV по сайтам XbaI/NotI для гена L и сайтам EcoRI/NotI для генов N и Р.
Амплификацию генов проводили с использованием специфических праймеров (см. ниже), а в качестве матрицы для амплификации использовали плазмиды pBS-L, pBS-N, pBS-P, соответственно.
Праймеры для клонирования N:
прямой: GCAAAGAATTCGCCGCCACCATGTCTGTTACAGTCAAGAGAATC
обратный: GAGGAGTGCGGCCGCTCATTTGTCAAATTCTGACTTAGC
Праймеры для клонирования Р:
прямой: GCAAAGAATTCGCCGCCACCATGGATAATCTCACAAAAGTTCG
обратный: GAGGAGTGCGGCCGCCTACAGAGAATATTTGACTCTCG
Праймеры для клонирования L (сборка из двух фрагментов):
прямой: GAATTCGCCGCCACCATGGAAGTCCACGATTTTGAGACC
обратный: GAGGAGTGCGGCCGCTTAATCTCTCCAAGAGTTTTCCTCGTG
прямой: GTGACCGGCGGCTCTAGAGCCTC
обратный: CTCAAAATCGTGGACTTCCATGGTGGCGGCGAATTCTTTGC
В результате были получены три хелперных плазмиды pCAG-VSVL (SEQ Id No.: 4), pCAG-VSVN (SEQ Id No.: 5), pCAG-VSVP (SEQ Id No.: 6), которые использовали для последующей трансфекции линии HEK293-TN (Фиг. 6).
Пример 3. Сборка VSV с использованием плазмиды, экспрессирующей Т7 полимеразу.
Для наработки rVSV и оценки уровня экспрессии GFP и, соответственно, сборки rVSV, на первом этапе использовали адгезионную клеточную линию HEK293TN и аналогичные условия трансфекции, описанные в Примере 1. Клетки культивировали на питательной среде DMEM+2mM L-Gln+2%FBS. В качестве трансфицирующего агента использовали PEL Для индукции экспрессии генов VSV использовали котрансфекцию с плазмидой pCAG-T7pol (cat# 59926) от Addgene.
В результате эксперимента после 48 часов обнаружили единичные группы GFP+ клеток (Фиг. 7), однако эффективность трансфекции была низкой для последующего использования кондиционированной культуральной жидкости на этапах амплификации rVSV - пассирования (минимальный порог, рекомендуемый по данным литературы - 20% GFP+ клеток).
Действительно, при переносе кондиционированной культуральной жидкости (трансдукции) мы не наблюдали положительного флуоресцентного сигнала, что говорит о низкой эффективности сборки rVSV. Для оптимизации процесса сборки решили получить клеточную линию, экспрессирующую Т7 полимеразу.
Пример 4. Получение стабильной клеточной линии, экспрессирующей Т7 полимеразу
Клонировали ген T7-RNApol в ретровирусный вектор pBabe-bleo (трансген устойчивости к зеоцину) по сайтам BamHI/Sal и нарабатывали вектор для трасфекции.
Для получения ретровируса, кодирующего T7-RNApol, клетки Phoenix-AMPHO - (CRL-3213 - АТСС) высевали на чашку 100 мм. Трансфекцию проводили при конфлюэнтности 70-80% предварительно полученной pBabe-bleo-T7-RNApol: 5 мкг плазмидной ДНК разведенной в 125 мкл Opti-MEM, смешивали с 25 мкг PEI в 125 мкл Opti-MEM (PEI:DNA=5:1). Через 12 часов после трансфекции меняли среду на 5,5 мл DMEM+10%FBS. Сбор культуральной жидкости с вирусом осуществляли каждые 12 часов
Клетки HEK293TN, высеянные в культуральный флакон с площадью поверхности 25 см2, заражали вирусной культуральной жидкости с ретровирусом в 3 приема, каждые 12 часов. Через 48 часов смена среды на DMEM+10%FBS+100мкг/мл зеоцин (Zeo). Селекция проводилась в течение недели на зеоцине 100 мкг/мл. Пересевали выжившие клетки в культуральный флакон с площадью поверхности 150 см2. Далее проводили скрининг отдельных колоний (через 2 недели после начала селекции). Для этого выделяли ДНК путем лизиса клеток колоний в 200 мкл буфера (200 мМ Tris-HCl, 250 мМ NaCl, 25 мМ EDTA, 0.5% SDS) и переосаждения фракции нуклеиновых кислот изопропанолом по стандартному протоколу. Проверку встройки трансгена T7-RNApol в ДНК клеток осуществляли методомом ПЦР (по 15 нг ДНК на ПЦР, праймеры:
t7rnapseq4f: Gaatcaggctgctggatacatg;
T7RNAP_sal_r: gttatctgtcgacctattacgcgaacgcgaagtccgactctaag;
отжиг на 55°C; элонгация 1 мин; ожидаемый ответ - 650 п.н.). Результаты скрининга на Фиг. 8.
Для последующего культивирования отобрали клоны 1, 4, 8 и 14 и выбрали клон 8 для дальнейшей работы. Проверка активности Т7 полимеразы в клоне 8 осуществлялась по уровню люминесценции люциферазы, экспрессирующейся с гена под Т7 промотором.
Для этого проводили временную трансфекцию клеток HEK293TN-T7 следующими экспрессионными векторами:
(1): плазмидой pEGFPN3 (отрицательный контроль + контроль трансфекции)
(2): плазмидой pS_5-Mod-LucF-3-Mod (люцифераза под Т7 промотором)
(3)): плазмидами pCAG-T7pol+pS_5-Mod-LucF-3-Mod (положительный контроль)
Оценивали уровень люминесценции после добавления субстрата к люциферазе (Фиг. 9).
Интенсивность биолюминесценции в линии HEK293TN-T7 была на уровне положительного контроля, что свидетельствует о том, что Т7 РНК-полимераза активна.
Таким образом, была получена клеточная линия HEK293TN-T7 (далее - HEK293-Т7) с устойчивостью к зеоцину, экспрессирующая активную Т7 полимеразу.
Пример 5. Подбор условий трансфекции клеточной линии, экспрессирующей Т7 полимеразу, для наработки rVSV.
Для проверки пригодности линии HEK293-Т7 к наработке rVSV проводили оптимизацию условий трансфекции: определение оптимального соотношения плазмид трансфекционной смеси, количества плазмидной ДНК на клетку, количества клеток, подбор трансфекционных агентов, соотношение ДНК: PEL а также влияния плазмид в смеси на получение rVSV, в том числе критичность наличия CAG-промотора в хелперных векторах. Несколько примеров составов трансфекционных смесей, использование которых не приводило к детекции GFP+ клеток на этапе трансфекции и не приводило к амплификации rVSV на последующих этапах, представлены ниже
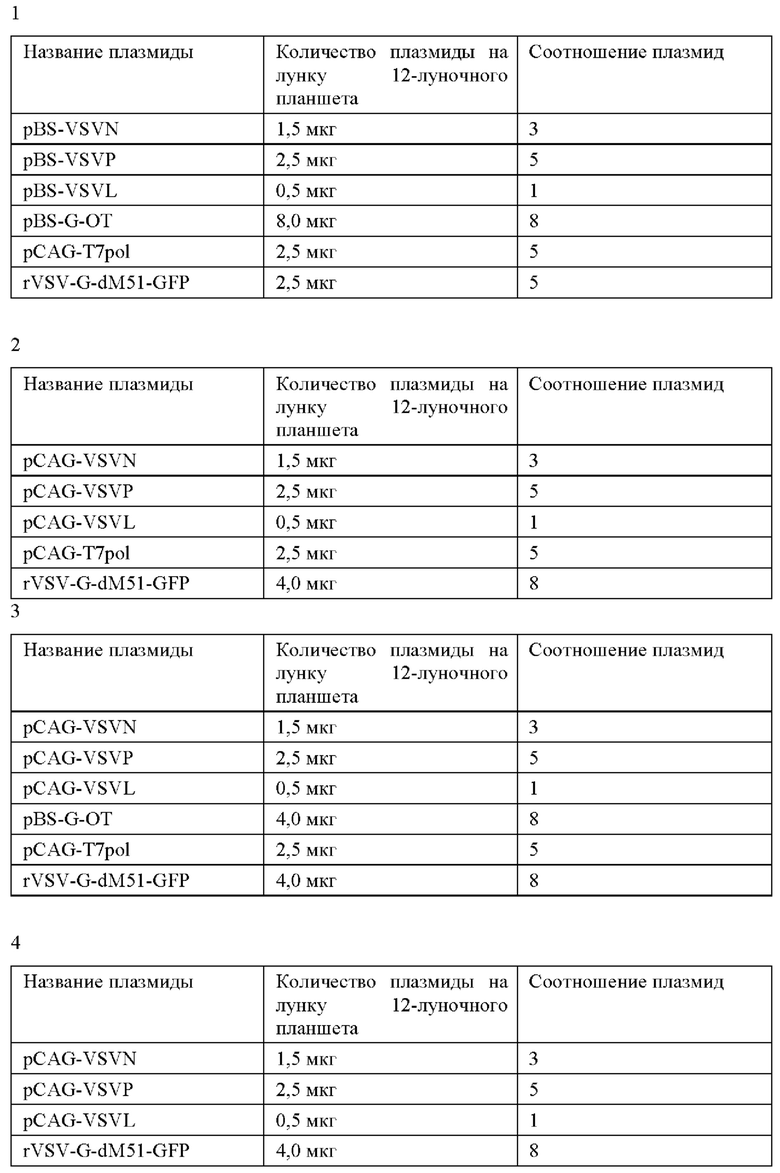
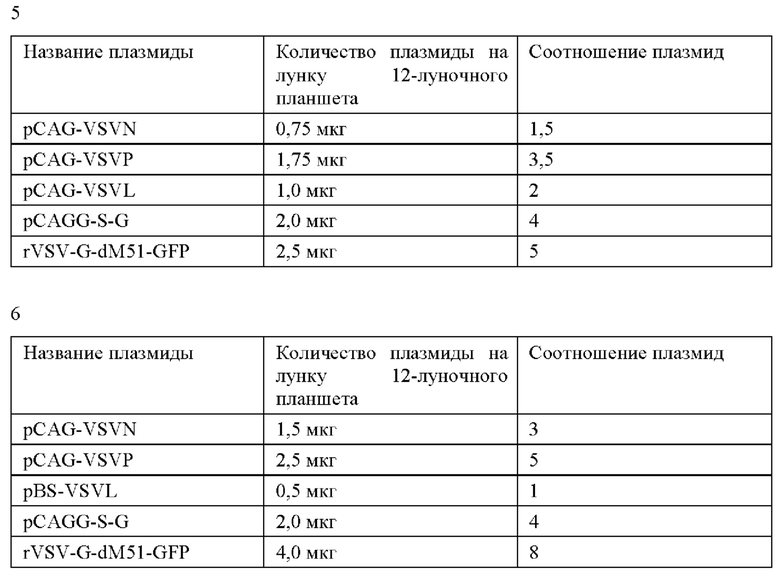
В результате был использованы условия проведения трансфекции, описанные в примере 1 как наиболее эффективные, но при этом получены 2 соотношения плазмид и общее количество ДНК (в мкг) в трансфекционной смеси, использование которых позволило стабильно получать от 50% и более GFP+ клеток (Фиг. 10) и в дальнейшем получать высокие титры rVSV на этапе амплификации.
Оптимизированное соотношение плазмид №1 (13 мкг на лунку 12 луночного планшета):
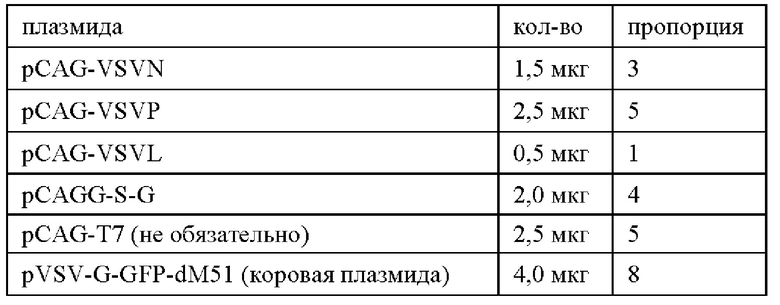
Оптимизированное соотношение плазмид №2 (6,75 мкг на лунку 12 луночного планшета)

За день до трансфекции во второй половине дня вносили по 350 000 клеток HEK293-Т7 в объеме среды 0,9 мл DMEM+10%FBS 0,9 мл в лунки 12-луночного планшета. На следующий день при достижении конфлюэнтности 75-90% за два часа до трансфекции меняли питательную среду на среду с более низким содержанием фетальной бычьей сыворотки (FBS, 2%). Для трансфекции на первом этапе для наработки rVSV-G-GFP-dM51 вирусных частиц использовали соотношение плазмид N:P:L:G: коровая 3:5:1:4:8 или 3:5:1:8:10. В случае, если дополнительно использовали плазмиду, кодирующую Т7 полимеразу, соотношение плазмид было М:Р:Ь:С:Т7:коровая 3:5:1:4:5:8 или 3:5:1:8:10:10, отличающееся от известных из уровня техники.
Приготовление трансфекционных смесей производили последовательным добавлением по отдельности в 2 центрифужные пробирки объемом 1,5 мл плазмид pCAG-VSVN, pCAG-VSVP, pCAG-VSVL, pCAGGS-G, pCAG-T7pol, pVSV-G-GFP-M51 в количестве указанном в таблицах, приведенных выше: с суммарной массой 13 мкг на лунку и PEI (65 мкг для соотношения РЕЕДНК 5:1). Если не использовали плазмиду pCAG-T7pol, суммарная масса ДНК была 10,5 мкг и количество PEI составляло 52,5 мкг. Далее в каждую пробирку вносили бессывороточную среду Opti-Mem из расчета 100 мкл на каждую лунку. После объединения смесей плазмид и PEI перемешивали смесь на вортексе и инкубировали при комнатной температуре в течение 10-15 минут (оптимально 13 минут). Готовые трансфицирующие смеси наносили по каплям на клетки в лунках с 0,9 мл питательной средой с низким содержанием FBS (2%). После этого планшеты переносили в инкубатор для культивирования. Режим культивирования: температура 37,0°С, влажность 90%, содержание СО2 5%. Утром следующего дня после трансфекции меняли среду на среду DMEM с высоким содержанием глюкозы (4,5 г/л) и низким содержанием FBS (2%). Через 72 часа после трансфекции переносили культуральную жидкость с клетками в стерильные центрифужные пробирки и при необходимости хранили при -80°С, либо использовали сразу для трансдукции.
Больший уровень флуоресценции наблюдали в образцах с дополнительной трансфекцией плазмидой pCAG-T7, несмотря на то, что клеточная линия стабильно экспрессирует Т7 полимеразу. Однако наличие или отсутствие плазмиды pCAG-T7 не влияло на эффективность последующей амплификации rVSV с точки зрения получаемого титра вируса.
Пример 6. Амплификация rVSV в клеточной линии BHK21 (этап трансдукции).
В качестве клеточной культуры для наработки rVSV на этапе амплификации была выбрана линия BHK21, т.к. после трансдукции rVSV, несущих ген GFP, в клетках этой линии наблюдался наибольший уровень флюоресцентного сигнала. Однако этот пример не ограничивает объем прав настоящего изобретения, поскольку специалист в данной области может взять клетки другой линии в зависимости от целей работы, имеющихся клеточных культур и опыта лаборатории. Наработку rVSV осуществляли по следующей схеме.
Перед вторым этапом наработки вирусных частиц (амплификация вирусных частиц, полученных на первом этапе) при необходимости проводили разморозку культуральной жидкости с клетками, полученную на первом этапе (Пример 5) и центрифугировали в течение 10 минут для избавления от дебриса клеток. После центрифугирования отбирали супернатант для последующей трансдукции и разбавляли его в два раза культуральной средой DMEM без сыворотки.
Для второго и последующих этапов наработки использовали клетки линии BHK21. Количество этапов наработки зависит от количества необходимого вируса (титр вируса в культуральной жидкости) для in vitro или in vivo экспериментов. За день до трансдукции вносили по 0,9 мл суспензии BHK21 в лунки 12-луночного планшета из расчета 250 000 клеток на лунку. На следующий день при достижении конфлюэнтности 55-75% во всех лунках снимали питательную среду, вносили по 500 мкл разбавленного в 2 раза супернатанта после 1 этапа сборки вирусных частиц и инкубировали 2 часа, затем добавляли по 500 мкл DMEM с 4% FBS. Через 72 ч культивирования снимали клетки с культуральной жидкостью и при необходимости хранили при 80°С, либо использовали сразу для повторной трансдукции или выделения, очистки, исследования вируса и пр.
Перед третьим этапом наработки вирусных частиц (амплификация вирусных частиц, полученных на втором этапе наработки) также при необходимости проводили разморозку культуральной жидкости с клетками и центрифугирование в течение 10 минут для избавления от дебриса клеток. Далее, также как перед вторым этапом наработки вируса, супернатант разбавляли в два раза культуральной средой DMEM без сыворотки. Для третьего этапа за день до трансдукции вносили по 5,0 мл суспензии BHK21 клеток в культуральные флаконы с площадью поверхности 25 см2 из расчета 1,5 млн клеток на флакон. На следующий день при достижении культурой клеток конфлюэнтности 55-75% удаляли питательную среду, вносили по 2,5 мл разбавленного в 2 раза супернатанта после 2 этапа наработки вирусных частиц и инкубировали 2 часа, затем добавляли по 2,5 мл DMEM с 4% FBS. Через 72 ч культивирования снимали клетки с культуральной жидкостью и при необходимости хранили при -80°С или использовали супернатанты для следующих этапов наработки или иных целей. На этом этапе отбирали образцы для подтверждения наличия вирусных частиц посредством методов PAGE и PCR.
После подтверждения наличия вирусных частиц проводили 4 этап наработки вирусных частиц при культивировании в культуральных флаконах с площадью поверхности 150 см2. Для этого за день до трансдукции вносили по 30,0 мл суспензии клеток линии BHK21 в культуральные флаконы с площадью поверхности 150 см2 из расчета 9,0 млн клеток на флакон. На следующий день при достижении конфлюэнтности 55-75% удаляли питательную среду, вносили по 15 мл разбавленного 2 раза супернатанта культуральной жидкости, полученного на 3 этапе наработки вирусных частиц, и инкубировали 2 часа, затем добавляли в каждый флакон по 15 мл среды для культивирования клеток DMEM с 4% FBS. Через 72 ч культивирования удаляли клетки с культуральной жидкостью, отбирали образцы для подтверждения наличия вирусных частиц посредством методов PAGE и PCR и при необходимости хранили при 80°С или использовали для последующих этапов наработки или других целей. На всех этапах сборки rVSV анализировали клеточные культуры с помощью флуоресцентного микроскопа и наблюдали высокий уровень флуоресценции через сутки после трансфекции/трансдукции и развитие цитопатического эффекта к 48 часам с угасанием уровня флуоресценции. Динамика развития цитопатического эффекта (цикл репликации - около суток) была сравнима с динамикой размножения вируса VSV дикого типа, описанной в работе [Hebert CG, DiNardo N, Evans ZL, Hart SJ, Hachmann AB. Rapid quantification of vesicular stomatitis virus in Vero cells using Laser Force Cytology. Vaccine. 2018 Oct l;36(41):6061-6069. doi: 10.1016/j.vaccine.2018.09.002].
Пример 7. Определение титра rVSV
Оценку титра вируса проводили по цитопатическому эффекту на клетки BHK21. Расчет титра (TCID50/ml) осуществляли по методу Рида-Менча, описанного в [Lei, С., Yang, J., Hu, J., & Sun, X. (2021). On the Calculation of TCID(50) for Quantitation of Virus Infectivity. Virol Sin, 36(1), 141-144. doi:10.1007/s12250-020-00230-5; Reed, L.J., & Muench, H. (1938). A simple method of estimating fifty per cent endpoints. American journal of epidemiology, 27(3), 493-497]. Коротко средняя инфекционная доза культуры ткани (TCID50) определяется как разведение вируса, необходимое для заражения 50% данной культуры клеток. Обратной величиной этого показателя является титр вируса в пересчете на инфекционную дозу на единицу объема.
Клетки BHK21 для анализа засевали в лунки 96-луночного планшета: 10 рядов по 6 лунок в каждом (всего 60 лунок). Для этого в каждую из лунок вносили по 25 000 клеток в 100 мкл среды DMEM+5% FBS с расчетом через 24 часа перед проведением эксперимента получить конфлюэнтность около 60-70%. 10 рядов соответствуют 9 рядам разведения вируса от 10-1 до 10-9 и 1 контрольному ряду, в каждом ряду по 6 повторностей. Готовили серию разведений супернатантов с rVSV, полученных на каждом этапе настоящего способа, в среде DMEM без FBS от 10-1 до 10-9, каждое следующее разведение получали из предыдущего разведением в 10 раз. Трансдукцию проводили, используя 50 мкл соответствующего разведения на лунку. Оставляли планшет инкубироваться при 37°С в течение 2 часов; через 2 часа в каждую лунку добавляли по 150 мкл заранее приготовленной среды DMEM+2,7% FBS для достижения конечной концентрации сыворотки 2% и оставляли планшет инкубироваться на 37°С в течение 72 часов. Через 72 часа под микроскопом фиксировали проявление цитопатического эффекта вируса в каждой лунке. Далее вычисляли TCID50/ml, используя стандартный протокол. Картину цитопатического действия (ЦПЭ) оформляли в виде схемы (Фиг. 11). Визуальный анализ лунок планшета и последующий расчет титра показал, что титр rVSV в супернатантах, полученных после наработки rVSV в культуральных флаконах площадью 150 см2, составлял 1,36⋅108 TCID50/мл.
Фотографии клеток BHK21 при наличии или отсутствии ЦПЭ приведены на Фиг. 12.
Пример 8. Анализ образцов VSV-dM51-GFP
Кондиционированную культуральную жидкостью клеток BHK21 после наработки rVSV концентрировали с помощью концентраторов (300 кДа, Vivaspin 2, Sartorius) по протоколу производителя и анализировали с помощью электрофореза в полиакриламидном геле в денатурирующих условиях по стандартной методике [Laemmli UK. Cleavage of Structural Proteins during the Assembly of the Head of Bacteriophage T4. Nature,1970; V.227, P. 680-685]. После окрашивания геля наблюдали полосы, молекулярная масса которых соответствуют пяти белкам rVSV. Дополнительно проверяли наличие в геноме rVSV гена GFP, используя для этого набор специфических праймеров к гену G и GFP (Фиг. 13, Фиг. 14, Фиг. 15).
Для этого 500 мкл культуральной жидкости клеток BHK21 - продуцентов рекомбинантного rVSV-G-GFP-dM51, обрабатывали протеинкиназой-К для высвобождения вирусной РНК. Далее РНК выделяли коммерческим набором «Лира+» (Биолабмикс, РФ). После выделения обрабатывали ДНКазой для удаления возможных остатков сторонней клеточной ДНК. Далее проводили обратную транскрипцию, используя специфичные праймеры, фланкирующие трансген GFP (ожидаемый ампликон 1175 п.н.):
1) 3'-конец гена g «vsvG_end_f»: CGGTCTCAAAATCGTGGACTTCC;
2) 5'-конец гена 1 «vsvL_start_r»: CATTCAAGACGCTGCTTCGCAAC
После получения кДНК проводили ПЦР с использованием этих же праймеров vsvG_end_f и vsvL_start_r (Taq-полимераза, денатурация 95°С 30 сек, отжиг 54°С 20 сек, элонгация 72°С 90 сек). Ампликоны анализировали на электрофорезе в агарозном геле. (Фиг. 13А, Фиг. 15)
В исследуемых образцах rVSV-G-GFP-dM51 детектировали полосу, соответствующую теоретически ожидаемому размеру фрагмента генома с геном GFP (Фиг. 13А), в то время как в контрольных образцах (Фиг. 15) детектировали фрагмент, соответствующий теоретически ожидаемому размеру фрагмента без гена GFP между G и L генами:

Пример 9. Наработка образцов рекомбинантных вирусов везикулярного стоматита в клеточной культуре HEK293, растущей на бессывороточной среде.
Проводили трансдукцию супернатантами rVSV-G-GFP-dM51, полученными способом, описанным в примере 6, суспензии клеток HEK293, растущих на бессывороточной среде (BalanCD HEK293, Irvine Scientific) и инкубировали 72 часа в СО2 инкубаторе, как описано выше. Через 72 часа после трансдукции центрифугировали клетки на 3000g, отбирали супернатант. Полученные супернатанты с этапов наработки фильтровали через шприцевые фильтры с диаметром пор 0,45 нм, концентрировали с помощью концентраторов (300 кДа, Vivaspin 2, Sartorius) по протоколу производителя в 100-200 раз и переводили в буфер PBS для хранения и анализа титра. Полученные концентраты титровали, определяли титр вируса (TCID50/мл) по методу Рида-Менча, как описано в Примере 7, и исследовали на белковом электрофорезе в полиакриламидном геле в денатурирующих условиях по стандартной методике [Laemmli UK. Cleavage of Structural Proteins during the Assembly of the Head of Bacteriophage T4. Nature, 1970; V.227, P. 680-685], подтверждая наличие в них полос, молекулярная масса которых соответствовала массе пяти белков VSV. При этом титр rVSV-G-GFP-dM51 в супернатанте до концентрирования достигал значения 2,9⋅107 TCID50/мл, а после концентрирования - более 108 TCID50/ml. Наработка rVSV в суспензионных культурах клеток млекопитающих может позволить легче масштабировать лабораторную технологию до объемов промышленных биореакторов и упростит процедуру наработки промышленных серий.
Пример 10. Получение рекомбинантных вирусов везикулярного стоматита VSV-mIL12-mGM-CSF, экспрессирующих химерный белок IL12-GMCSF.
Вирус VSV-mIL12-mGM-CSF получали, как описано в Примерах 5 и 6, при этом в качестве коровой плазмиды использовали плазмиду pVSV-G-dM51-mIL12-mGMCSF, схема которой приведена на Фиг. 16 или 17. Коровая плазмида отличается тем, что в качестве трансгена содержит последовательность SEQ Id NO.: 7 или 8, кодирующую химерный белок IL12-GMCSF, в котором IL12 и GMCSF соединены коротким или длинным линкером соответственно. Титр вируса составил 9,3⋅105 TCID50/мл. Полученные рекомбинантные вирусы везикулярного стоматита демонстрируют стабильную экспрессию химерного белка, обладающего цитокиновой активностью. При этом химерные белки проявляют свою функциональность, демонстрируя способность активировать компетентные клетки in vitro. Также, in vivo показано, что рекомбинантный вирус везикулярного стоматита VSV-mIL12-mGM-CSF обладает выраженной противоопухолевой активностью в животной модели опухолевого ксенотрансплантата. Онколитическая способность полученного рекомбинантного вируса везикулярного стоматита превышала онколитическую способность аналогичного вируса, не экспрессирующего химерный IL12-GMCSF, в 1000 раз. Препарат VSV-mIL12-mGM-CSF обладал терапевтическим эффектом в 1000-кратно меньшей дозе из расчета титров по TCID50.
Таким образом, способ по настоящему изобретению позволяет получать рекомбинантный вирус везикулярного стоматита в титре, сравнимом с инфекционным титром вируса везикулярного стоматита дикого типа. При этом способ по настоящему изобретению не требует использования дополнительных живых вирусов. rVSV, полученный способом по настоящему изобретению, также не отличается от вируса дикого типа по динамике накопления в клетках [Hebert CG, DiNardo N, Evans ZL, et al. Rapid quantification of vesicular stomatitis virus in Vero cells using Laser Force Cytology. Vaccine. 2018 Oct 1;36(41):6061-6069. doi: 10.1016/j.vaccine.2018.09.002].
Все публикации, патенты и заявки на патенты включены в настоящий документ посредством ссылки. Хотя в вышеприведенном описании это изобретение было описано в отношении некоторых предпочтительных вариантов его осуществления, и многие детали были изложены в целях иллюстрации, для специалистов в данной области техники будет очевидно, что изобретение допускает дополнительные варианты осуществления и что некоторые детали, описанные в данном документе, могут значительно изменяться без отклонения от сущности изобретения.
Использование терминов в единственном числе в контексте описания изобретения должно толковаться как охватывающее как единственное, так и множественное число, если иное не указано в данном документе или явно не противоречит контексту. Термины «состоящий из», «имеющий», «включающий» и «содержащий» следует толковать как неограничивающие термины, т.е. означающие «включая, но не ограничиваясь», если не указано иное. Перечисление диапазонов значений в данном документе просто предназначено для использования в качестве сокращенного способа индивидуальной ссылки на каждое отдельное значение, попадающее в этот диапазон, если здесь не указано иное, и каждое отдельное значение включено в спецификацию, как если бы оно было отдельно изложено в данном документе. Все способы, описанные в данном документе, могут выполняться в любом подходящем порядке, если иное не указано в данном документе или иным образом явно не противоречит контексту. Использование любых и всех примеров или иллюстративного языка (например, «такой как»), представленных в данном документе, предназначено просто для лучшего описания изобретения и не налагает ограничения на объем изобретения, если иное не заявлено. Никакие формулировки в описании не следует истолковывать как указывающие на какой-либо не заявленный элемент как существенный для практического применения изобретения.
Здесь описаны варианты осуществления этого изобретения, включая лучший из известных изобретателям способа осуществления изобретения. Разновидности этих вариантов осуществления могут стать очевидными для специалистов в данной области техники после прочтения предшествующего описания. Авторы ожидают, что квалифицированные специалисты будут использовать такие варианты в зависимости от обстоятельств, и авторы предполагают, что изобретение будет реализовано на практике иначе, чем конкретно описано в данном документе. Соответственно, это изобретение включает в себя все модификации и эквиваленты признаков, изложенных в прилагаемой формуле изобретения, как это разрешено действующим законодательством. Более того, любая комбинация вышеописанных признаков во всех их возможных вариациях охватывается изобретением, если иное не указано в данном документе или иным образом явно не противоречит контексту.
Заявитель просит рассмотреть представленные материалы заявки «Способ получения рекомбинантного вируса везикулярного стоматита» на предмет выдачи патента на изобретение.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="VSV способ.xml"
softwareName="WIPO Sequence" softwareVersion="2.2.0"
productionDate="2022-12-27">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2022134626</ApplicationNumberText>
<FilingDate>2022-12-27</FilingDate>
</ApplicationIdentification>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2022134626</ApplicationNumberText>
<FilingDate>2022-12-27</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Автономная некоммерческая
образовательная организация высшего образования
"Научно-Технологический Университет
"Сириус"</ApplicantName>
<ApplicantNameLatin>Sirius University of Science and
Technology</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ получения рекомбинантного
вируса везикулярного стоматита</InventionTitle>
<SequenceTotalQuantity>8</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>422</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..422</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Vesiculovirus
indiana</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MSVTVKRIIDNTVIVPKLPANEDPVEYPADYFRKSKEIPLYINTTKSLS
DLRGYVYQGLKSGNVSIIHVNSYLYGALKDIRGKLDKDWSSFGINIGKAGDTIGIFDLVSLKALDGVLPD
GVSDASRTSADDKWLPLYLLGLYRVGRTQMPEYRKKLMDGLTNQCKMINEQFEPLVPEGRDIFDVWGNDS
NYTKIVAAVDMFFHMFKKHECASFRYGTIVSRFKDCAALATFGHLCKITGMSTEDVTTWILNREVADEMV
QMMLPGQEIDKADSYMPYLIDFGLSSKSPYSSVKNPAFHFWGQLTALLLRSTRARNARQPDDIEYTSLTT
AGLLYAYAVGSSADLAQQFCVGDNKYTPDDSTGGLTTNAPPQGRDVVEWLGWFEDQNRKPTPDMMQYAKR
AVMSLQGLREKTIGKYAKSEFDK</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>265</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..265</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Vesiculovirus
indiana</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MDNLTKVREYLKSYSRLDQAVGEIDEIEAQRAEKSNYELFQEDGVEEHT
KPSYFQAADDSDTESEPEIEDNQGLYAQDPEAEQVEGFIQGPLDDYADEEVDVVFTSDWKQPELESDEHG
KTLRLTSPEGLSGEQKSQWLSTIKAVVQSAKYWNLAECTFEASGEGVIMKERQITPDVYKVTPVMNTHPS
QSEAVSDVWSLSKTSMTFQPKKASLQPLTISLDELFSSRGEFISVGGDGRMSHKEAILLGLRYKKLYNQA
RVKYSL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>2109</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..2109</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Vesiculovirus
indiana</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MEVHDFETDEFNDFNEDDYATREFLNPDERMTYLNHADYNLNSPLISDD
IDNLIRKFNSLPIPSMWDSKNWDGVLEMLTSCQANPISTSQMHKWMGSWLMSDNHDASQGYSFLHEVDKE
AEITFDVVETFIRGWGNKPIEYIKKERWTDSFKILAYLCQKFLDLHKLTLILNAVSEVELLNLARTFKGK
VRRSSHGTNICRIRVPSLGPTFISEGWAYFKKLDILMDRNFLLMVKDVIIGRMQTVLSMVCRIDNLFSEQ
DIFSLLNIYRIGDKIVERQGNFSYDLIKMVEPICNLKLMKLARESRPLVPQFPHFENHIKTSVDEGAKID
RGIRFLHDQIMSVKTVDLTLVIYGSFRHWGHPFIDYYTGLEKLHSQVTMKKDIDVSYAKALASDLARIVL
FQQFNDHKKWFVNGDLLPHDHPFKSHVKENTWPTAAQVQDFGDKWHELPLIKCFEIPDLLDPSIIYSDKS
HSMNRSEVLKHVRMNPNTPIPSKKVLQTMLDTKATNWKEFLKEIDEKGLDDDDLIIGLKGKERELKLAGR
FFSLMSWKLREYFVITEYLIKTHFVPMFKGLTMADDLTAVIKKMLDSSSGQGLKSYEAICIANHIDYEKW
NNHQRKLSNGPVFRVMGQFLGYPSLIERTHEFFEKSLIYYNGRPDLMRVHNNTLINSTSQRVCWQGQEGG
LEGLRQKGWTILNLLVIQREAKIRNTAVKVLAQGDNQVICTQYKTKKSRNVVELQGALNQMVSNNEKIMT
AIKIGTGKLGLLINDDETMQSADYLNYGKIPIFRGVIRGLETKRWSRVTCVTNDQIPTCANIMSSVSTNA
LTVAHFAENPINAMIQYNYFGTFARLLLMMHDPALRQSLYEVQDKIPGLHSSTFKYAMLYLDPSIGGVSG
MSLSRFLIRAFPDPVTESLSFWRFIHVHARSEHLKEMSAVFGNPEIAKFRITHIDKLVEDPTSLNIAMGM
SPANLLKTEVKKCLIESRQTIRNQVIKDATIYLYHEEDRLRSFLWSINPLFPRFLSEFKSGTFLGVADGL
ISLFQNSRTIRNSFKKKYHRELDDLIVRSEVSSLTHLGKLHLRRGSCKMWTCSATHADTLRYKSWGRTVI
GTTVPHPLEMLGPQHRKETPCAPCNTSGFNYVSVHCPDGIHDVFSSRGPLPAYLGSKTSESTSILQPWER
ESKVPLIKRATRLRDAISWFVEPDSKLAMTILSNIHSLTGEEWTKRQHGFKRTGSALHRFSTSRMSHGGF
ASQSTAALTRLMATTDTMRDLGDQNFDFLFQATLLYAQITTTVARDGWITSCTDHYHIACKSCLRPIEEI
TLDSSMDYTPPDVSHVLKTWRNGEGSWGQEIKQIYPLEGNWKNLAPAEQSYQVGRCIGFLYGDLAYRKST
HAEDSSLFPLSIQGRIRGRGFLKGLLDGLMRASCCQVIHRRSLAHLKRPANAVYGGLIYLIDKLSVSPPF
LSLTRSGPIRDELETIPHKIPTSYPTSNRDMGVIVRNYFKYQCRLIEKGKYRSHYSQLWLFSDVLSIDFI
GPFSISTTLLQILYKPFLSGKDKNELRELANLSSLLRSGEGWEDIHVKFFTKDILLCPEEIRHACKFGIA
KDNNKDMSYPPWGRESRGTITTIPVYYTTTPYPKMLEMPPRIQNPLLSGIRLGQLPTGAHYKIRSILHGM
GIHYRDFLSCGDGSGGMTAALLRENVHSRGIFNSLLELSGSVMRGASPEPPSALETLGGDKSRCVNGETC
WEYPSDLCDPRTWDYFLRLKAGLGLQIDLIVMDMEVRDSSTSLKIETNVRNYVHRILDEQGVLIYKTYGT
YICESEKNAVTILGPMFKTVDLVQTEFSSSQTSEVYMVCKGLKKLIDEPNPDWSSINESWKNLYAFQSSE
QEFARAKKVSTYFTLTGIPSQFIPDPFVNIETMLQIFGVPTGVSHAAALKSSDRPADLLTISLFYMAIIS
YYNINHIRVGPIPPNPPSDGIAQNVGIAITGISFWLSLMEKDIPLYQQCLAVIQQSFPIRWEAVSVKGGY
KQKWSTRGDGLPKDTRISDSLAPIGNWIRSLELVRNQVRLNPFNEILFNQLCRTVDNHLKWSNLRRNTGM
IEWINRRISKEDRSILMLKSDLHEENSWRD</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>11130</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11130</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtcgacattgattattgactagttattaatagtaatcaattacggggt
cattagttcatagcccatatatggagttccgcgttacataacttacggtaaatggcccgcctggctgacc
gcccaacgacccccgcccattgacgtcaataatgacgtatgttcccatagtaacgccaatagggactttc
cattgacgtcaatgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgc
caagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagtacatgacctt
atgggactttcctacttggcagtacatctacgtattagtcatcgctattaccatggtcgaggtgagcccc
acgttctgcttcactctccccatctcccccccctccccacccccaattttgtatttatttattttttaat
tattttgtgcagcgatgggggcggggggggggggggcgcgcgccaggcggggcggggcggggcgaggggc
ggggcggggcgaggcggagaggtgcggcggcagccaatcagagcggcgcgctccgaaagtttccttttat
ggcgaggcggcggcggcggcggccctataaaaagcgaagcgcgcggcgggcgggagtcgctgcgttgcct
tcgccccgtgccccgctccgcgccgcctcgcgccgcccgccccggctctgactgaccgcgttactcccac
aggtgagcgggcgggacggcccttctcctccgggctgtaattagcgcttggtttaatgacggctcgtttc
ttttctgtggctgcgtgaaagccttaaagggctccgggagggccctttgtgcgggggggagcggctcggg
gggtgcgtgcgtgtgtgtgtgcgtggggagcgccgcgtgcggcccgcgctgcccggcggctgtgagcgct
gcgggcgcggcgcggggctttgtgcgctccgcgtgtgcgcgaggggagcgcggccgggggcggtgccccg
cggtgcgggggggctgcgaggggaacaaaggctgcgtgcggggtgtgtgcgtgggggggtgagcaggggg
tgtgggcgcggcggtcgggctgtaacccccccctgcacccccctccccgagttgctgagcacggcccggc
ttcgggtgcggggctccgtgcggggcgtggcgcggggctcgccgtgccgggcggggggtggcggcaggtg
ggggtgccgggcggggcggggccgcctcgggccggggagggctcgggggaggggcgcggcggccccggag
cgccggcggctgtcgaggcgcggcgagccgcagccattgccttttatggtaatcgtgcgagagggcgcag
ggacttcctttgtcccaaatctgtgcggagccgaaatctgggaggcgccgccgcaccccctctagcgggc
gcggggcgaagcggtgcggcgccggcaggaaggaaatgggcggggagggccttcgtgcgtcgccgcgccg
ccgtccccttctccctctccagcctcggggctgtccgcggggggacggctgccttcgggggggacggggc
agggcggggttcggcttctggcgtgtgaccggcggctctagagcctctgctaaccatgttcatgccttct
tctttttcctacagctcctgggcaacgtgctggttattgtgctgtctcatcattttggcaaagaattcgc
cgccaccatggaagtccacgattttgagaccgacgagttcaatgatttcaatgaagatgactatgccaca
agagaattcctgaatcccgatgagcgcatgacgtacttgaatcatgctgattacaatttgaattctcctc
taattagtgatgatattgacaatttgatcaggaaattcaattctcttccgattccctcgatgtgggatag
taagaactgggatggagttcttgagatgttaacatcatgtcaagccaatcccatctcaacatctcagatg
cataaatggatgggaagttggttaatgtctgataatcatgatgccagtcaagggtatagttttttacatg
aagtggacaaagaggcagaaataacatttgacgtggtggagaccttcatccgcggctggggcaacaaacc
aattgaatacatcaaaaaggaaagatggactgactcattcaaaattctcgcttatttgtgtcaaaagttt
ttggacttacacaagttgacattaatcttaaatgctgtctctgaggtggaattgctcaacttggcgagga
ctttcaaaggcaaagtcagaagaagttctcatggaacgaacatatgcaggattagggttcccagcttggg
tcctacttttatttcagaaggatgggcttacttcaagaaacttgatattctaatggaccgaaactttctg
ttaatggtcaaagatgtgattatagggaggatgcaaacggtgctatccatggtatgtagaatagacaacc
tgttctcagagcaagacatcttctcccttctaaatatctacagaattggagataaaattgtggagaggca
gggaaatttttcttatgacttgattaaaatggtggaaccgatatgcaacttgaagctgatgaaattagca
agagaatcaaggcctttagtcccacaattccctcattttgaaaatcatatcaagacttctgttgatgaag
gggcaaaaattgaccgaggtataagattcctccatgatcagataatgagtgtgaaaacagtggatctcac
actggtgatttatggatcgttcagacattggggtcatccttttatagattattacactggactagaaaaa
ttacattcccaagtaaccatgaagaaagatattgatgtgtcatatgcaaaagcacttgcaagtgatttag
ctcggattgttctatttcaacagttcaatgatcataaaaagtggttcgtgaatggagacttgctccctca
tgatcatccctttaaaagtcatgttaaagaaaatacatggcccacagctgctcaagttcaagattttgga
gataaatggcatgaacttccgctgattaaatgttttgaaatacccgacttactagacccatcgataatat
actctgacaaaagtcattcaatgaataggtcagaggtgttgaaacatgtccgaatgaatccgaacactcc
tatccctagtaaaaaggtgttgcagactatgttggacacaaaggctaccaattggaaagaatttcttaaa
gagattgatgagaagggcttagatgatgatgatctaattattggtcttaaaggaaaggagagggaactga
agttggcaggtagatttttctccctaatgtcttggaaattgcgagaatactttgtaattaccgaatattt
gataaagactcatttcgtccctatgtttaaaggcctgacaatggcggacgatctaactgcagtcattaaa
aagatgttagattcctcatccggccaaggattgaagtcatatgaggcaatttgcatagccaatcacattg
attacgaaaaatggaataaccaccaaaggaagttatcaaacggcccagtgttccgagttatgggccagtt
cttaggttatccatccttaatcgagagaactcatgaattttttgagaaaagtcttatatactacaatgga
agaccagacttgatgcgtgttcacaacaacacactgatcaattcaacctcccaacgagtttgttggcaag
gacaagagggtggactggaaggtctacggcaaaaaggatggactatcctcaatctactggttattcaaag
agaggctaaaatcagaaacactgctgtcaaagtcttggcacaaggtgataatcaagttatttgcacacag
tataaaacgaagaaatcgagaaacgttgtagaattacagggtgctctcaatcaaatggtttctaataatg
agaaaattatgactgcaatcaaaatagggacagggaagttaggacttttgataaatgacgatgagactat
gcaatctgcagattacttgaattatggaaaaataccgattttccgtggagtgattagagggttagagacc
aagagatggtcacgagtgacttgtgtcaccaatgaccaaatacccacttgtgctaatataatgagctcag
tttccacaaatgctctcaccgtagctcattttgctgagaacccaatcaatgccatgatacagtacaatta
ttttgggacatttgctagactcttgttgatgatgcatgatcctgctcttcgtcaatcattgtatgaagtt
caagataagataccgggcttgcacagttctactttcaaatacgccatgttgtatttggacccttccattg
gaggagtgtcgggcatgtctttgtccaggtttttgattagagccttcccagatcccgtaacagaaagtct
ctcattctggagattcatccatgtacatgctcgaagtgagcatctgaaggagatgagtgcagtatttgga
aaccccgagatagccaagtttcgaataactcacatagacaagctagtagaagatccaacctctctgaaca
tcgctatgggaatgagtccagcgaacttgttaaagactgaggttaaaaaatgcttaatcgaatcaagaca
aaccatcaggaaccaggtgattaaggatgcaaccatatatttgtatcatgaagaggatcggctcagaagt
ttcttatggtcaataaatcctctgttccctagatttttaagtgaattcaaatcaggcacttttttgggag
tcgcagacgggctcatcagtctatttcaaaattctcgtactattcggaactcctttaagaaaaagtatca
tagggaattggatgatttgattgtgaggagtgaggtatcctctttgacacatttagggaaacttcatttg
agaaggggatcatgtaaaatgtggacatgttcagctactcatgctgacacattaagatacaaatcctggg
gccgtacagttattgggacaactgtaccccatccattagaaatgttgggtccacaacatcgaaaagagac
tccttgtgcaccatgtaacacatcagggttcaattatgtttctgtgcattgtccagacgggatccatgac
gtctttagttcacggggaccattgcctgcttatctagggtctaaaacatctgaatctacatctattttgc
agccttgggaaagggaaagcaaagtcccactgattaaaagagctacacgtcttagagatgctatctcttg
gtttgttgaacccgactctaaactagcaatgactatactttctaacatccactctttaacaggcgaagaa
tggaccaaaaggcagcatgggttcaaaagaacagggtctgcccttcataggttttcgacatctcggatga
gccatggtgggttcgcatctcagagcactgcagcattgaccaggttgatggcaactacagacaccatgag
ggatctgggagatcagaatttcgactttttattccaagcaacgttgctctatgctcaaattaccaccact
gttgcaagagacggatggatcaccagttgtacagatcattatcatattgcctgtaagtcctgtttgagac
ccatagaagagatcaccctggactcaagtatggactacacgcccccagatgtatcccatgtgctgaagac
atggaggaatggggaaggttcgtggggacaagagataaaacagatctatcctttagaagggaattggaag
aatttagcacctgctgagcaatcctatcaagtcggcagatgtataggttttctatatggagacttggcgt
atagaaaatctactcatgccgaggacagttctctatttcctctatctatacaaggtcgtattagaggtcg
aggtttcttaaaagggttgctagacggattaatgagagcaagttgctgccaagtaatacaccggagaagt
ctggctcatttgaagaggccggccaacgcagtgtacggaggtttgatttacttgattgataaattgagtg
tatcacctccattcctttctcttactagatcaggacctattagagacgaattagaaacgattccccacaa
gatcccaacctcctatccgacaagcaaccgtgatatgggggtgattgtcagaaattacttcaaataccaa
tgccgtctaattgaaaagggaaaatacagatcacattattcacaattatggttattctcagatgtcttat
ccatagacttcattggaccattctctatttccaccaccctcttgcaaatcctatacaagccatttttatc
tgggaaagataagaatgagttgagagagctggcaaatctttcttcattgctaagatcaggagaggggtgg
gaagacatacatgtgaaattcttcaccaaggacatattattgtgtccagaggaaatcagacatgcttgca
agttcgggattgctaaggataataataaagacatgagctatcccccttggggaagggaatccagagggac
aattacaacaatccctgtttattatacgaccaccccttacccaaagatgctagagatgcctccaagaatc
caaaatcccctgctgtccggaatcaggttgggccaattaccaactggcgctcattataaaattcggagta
tattacatggaatgggaatccattacagggacttcttgagttgtggagacggctccggagggatgactgc
tgcattactacgagaaaatgtgcatagcagaggaatattcaatagtctgttagaattatcagggtcagtc
atgcgaggcgcctctcctgagccccccagtgccctagaaactttaggaggagataaatcgagatgtgtaa
atggtgaaacatgttgggaatatccatctgacttatgtgacccaaggacttgggactatttcctccgact
caaagcaggcttggggcttcaaattgatttaattgtaatggatatggaagttcgggattcttctactagc
ctgaaaattgagacgaatgttagaaattatgtgcaccggattttggatgagcaaggagttttaatctaca
agacttatggaacatatatttgtgagagcgaaaagaatgcagtaacaatccttggtcccatgttcaagac
ggtcgacttagttcaaacagaatttagtagttctcaaacgtctgaagtatatatggtatgtaaaggtttg
aagaaattaatcgatgaacccaatcccgattggtcttccatcaatgaatcctggaaaaacctgtacgcat
tccagtcatcagaacaggaatttgccagagcaaagaaggttagtacatactttaccttgacaggtattcc
ctcccaattcattcctgatccttttgtaaacattgagactatgctacaaatattcggagtacccacgggt
gtgtctcatgcggctgccttaaaatcatctgatagacctgcagatttattgaccattagccttttttata
tggcgattatatcgtattataacatcaatcatatcagagtaggaccgatacctccgaaccccccatcaga
tggaattgcacaaaatgtggggatcgctataactggtataagcttttggctgagtttgatggagaaagac
attccactatatcaacagtgtttagcagttatccagcaatcattcccgattaggtgggaggctgtttcag
taaaaggaggatacaagcagaagtggagtactagaggtgatgggctcccaaaagatacccgaatttcaga
ctccttggccccaatcgggaactggatcagatctctggaattggtccgaaaccaagttcgtctaaatcca
ttcaatgagatcttgttcaatcagctatgtcgtacagtggataatcatttgaaatggtcaaatttgcgaa
gaaacacaggaatgattgaatggatcaatagacgaatttcaaaagaagaccggtctatactgatgttgaa
gagtgacctacacgaggaaaactcttggagagattaagcggccgcactcctcaggtgcaggctgcctatc
agaaggtggtggctggtgtggccaatgccctggctcacaaataccactgagatctttttccctctgccaa
aaattatggggacatcatgaagccccttgagcatctgacttctggctaataaaggaaatttattttcatt
gcaatagtgtgttggaattttttgtgtctctcactcggaaggacatatgggagggcaaatcatttaaaac
atcagaatgagtatttggtttagagtttggcaacatatgcccatatgctggctgccatgaacaaaggttg
gctataaagaggtcatcagtatatgaaacagccccctgctgtccattccttattccatagaaaagccttg
acttgaggttagattttttttatattttgttttgtgttatttttttctttaacatccctaaaattttcct
tacatgttttactagccagatttttcctcctctcctgactactcccagtcatagctgtccctcttctctt
atggagatccctcgacctgcagcccaagcttggcgtaatcatggtcatagctgtttcctgtgtgaaattg
ttatccgctcacaattccacacaacatacgagccggaagcataaagtgtaaagcctggggtgcctaatga
gtgagctaactcacattaattgcgttgcgctcactgcccgctttccagtcgggaaacctgtcgtgccagc
ggatccgcatctcaattagtcagcaaccatagtcccgcccctaactccgcccatcccgcccctaactccg
cccagttccgcccattctccgccccatggctgactaattttttttatttatgcagaggccgaggccgcct
cggcctctgagctattccagaagtagtgaggaggcttttttggaggcctaggcttttgcaaaaagctaac
ttgtttattgcagcttataatggttacaaataaagcaatagcatcacaaatttcacaaataaagcatttt
tttcactgcattctagttgtggtttgtccaaactcatcaatgtatcttatcatgtctggatccgctgcat
taatgaatcggccaacgcgcggggagaggcggtttgcgtattgggcgctcttccgcttcctcgctcactg
actcgctgcgctcggtcgttcggctgcggcgagcggtatcagctcactcaaaggcggtaatacggttatc
cacagaatcaggggataacgcaggaaagaacatgtgagcaaaaggccagcaaaaggccaggaaccgtaaa
aaggccgcgttgctggcgtttttccataggctccgcccccctgacgagcatcacaaaaatcgacgctcaa
gtcagaggtggcgaaacccgacaggactataaagataccaggcgtttccccctggaagctccctcgtgcg
ctctcctgttccgaccctgccgcttaccggatacctgtccgcctttctcccttcgggaagcgtggcgctt
tctcatagctcacgctgtaggtatctcagttcggtgtaggtcgttcgctccaagctgggctgtgtgcacg
aaccccccgttcagcccgaccgctgcgccttatccggtaactatcgtcttgagtccaacccggtaagaca
cgacttatcgccactggcagcagccactggtaacaggattagcagagcgaggtatgtaggcggtgctaca
gagttcttgaagtggtggcctaactacggctacactagaagaacagtatttggtatctgcgctctgctga
agccagttaccttcggaaaaagagttggtagctcttgatccggcaaacaaaccaccgctggtagcggtgg
tttttttgtttgcaagcagcagattacgcgcagaaaaaaaggatctcaagaagatcctttgatcttttct
acggggtctgacgctcagtggaacgaaaactcacgttaagggattttggtcatgagattatcaaaaagga
tcttcacctagatccttttaaattaaaaatgaagttttaaatcaatctaaagtatatatgagtaaacttg
gtctgacagttaccaatgcttaatcagtgaggcacctatctcagcgatctgtctatttcgttcatccata
gttgcctgactccccgtcgtgtagataactacgatacgggagggcttaccatctggccccagtgctgcaa
tgataccgcgagacccacgctcaccggctccagatttatcagcaataaaccagccagccggaagggccga
gcgcagaagtggtcctgcaactttatccgcctccatccagtctattaattgttgccgggaagctagagta
agtagttcgccagttaatagtttgcgcaacgttgttgccattgctacaggcatcgtggtgtcacgctcgt
cgtttggtatggcttcattcagctccggttcccaacgatcaaggcgagttacatgatcccccatgttgtg
caaaaaagcggttagctccttcggtcctccgatcgttgtcagaagtaagttggccgcagtgttatcactc
atggttatggcagcactgcataattctcttactgtcatgccatccgtaagatgcttttctgtgactggtg
agtactcaaccaagtcattctgagaatagtgtatgcggcgaccgagttgctcttgcccggcgtcaatacg
ggataataccgcgccacatagcagaactttaaaagtgctcatcattggaaaacgttcttcggggcgaaaa
ctctcaaggatcttaccgctgttgagatccagttcgatgtaacccactcgtgcacccaactgatcttcag
catcttttactttcaccagcgtttctgggtgagcaaaaacaggaaggcaaaatgccgcaaaaaagggaat
aagggcgacacggaaatgttgaatactcatactcttcctttttcaatattattgaagcatttatcagggt
tattgtctcatgagcggatacatatttgaatgtatttagaaaaataaacaaataggggttccgcgcacat
ttccccgaaaagtgccacctg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>6069</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..6069</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtcgacattgattattgactagttattaatagtaatcaattacggggt
cattagttcatagcccatatatggagttccgcgttacataacttacggtaaatggcccgcctggctgacc
gcccaacgacccccgcccattgacgtcaataatgacgtatgttcccatagtaacgccaatagggactttc
cattgacgtcaatgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgc
caagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagtacatgacctt
atgggactttcctacttggcagtacatctacgtattagtcatcgctattaccatggtcgaggtgagcccc
acgttctgcttcactctccccatctcccccccctccccacccccaattttgtatttatttattttttaat
tattttgtgcagcgatgggggcggggggggggggggcgcgcgccaggcggggcggggcggggcgaggggc
ggggcggggcgaggcggagaggtgcggcggcagccaatcagagcggcgcgctccgaaagtttccttttat
ggcgaggcggcggcggcggcggccctataaaaagcgaagcgcgcggcgggcgggagtcgctgcgttgcct
tcgccccgtgccccgctccgcgccgcctcgcgccgcccgccccggctctgactgaccgcgttactcccac
aggtgagcgggcgggacggcccttctcctccgggctgtaattagcgcttggtttaatgacggctcgtttc
ttttctgtggctgcgtgaaagccttaaagggctccgggagggccctttgtgcgggggggagcggctcggg
gggtgcgtgcgtgtgtgtgtgcgtggggagcgccgcgtgcggcccgcgctgcccggcggctgtgagcgct
gcgggcgcggcgcggggctttgtgcgctccgcgtgtgcgcgaggggagcgcggccgggggcggtgccccg
cggtgcgggggggctgcgaggggaacaaaggctgcgtgcggggtgtgtgcgtgggggggtgagcaggggg
tgtgggcgcggcggtcgggctgtaacccccccctgcacccccctccccgagttgctgagcacggcccggc
ttcgggtgcggggctccgtgcggggcgtggcgcggggctcgccgtgccgggcggggggtggcggcaggtg
ggggtgccgggcggggcggggccgcctcgggccggggagggctcgggggaggggcgcggcggccccggag
cgccggcggctgtcgaggcgcggcgagccgcagccattgccttttatggtaatcgtgcgagagggcgcag
ggacttcctttgtcccaaatctgtgcggagccgaaatctgggaggcgccgccgcaccccctctagcgggc
gcggggcgaagcggtgcggcgccggcaggaaggaaatgggcggggagggccttcgtgcgtcgccgcgccg
ccgtccccttctccctctccagcctcggggctgtccgcggggggacggctgccttcgggggggacggggc
agggcggggttcggcttctggcgtgtgaccggcggctctagagcctctgctaaccatgttcatgccttct
tctttttcctacagctcctgggcaacgtgctggttattgtgctgtctcatcattttggcaaagaattcgc
cgccaccatgtctgttacagtcaagagaatcattgacaacacagtcatagttccaaaacttcctgcaaat
gaggatccagtggaatacccggcagattacttcagaaaatcaaaggagattcctctttacatcaatacta
caaaaagtttgtcagatctaagaggatatgtctaccaaggcctcaaatccggaaatgtatcaatcataca
tgtcaacagctacttgtatggagcattaaaggacatccggggtaagttggataaagattggtcaagtttc
ggaataaacatcgggaaagcaggggatacaatcggaatatttgaccttgtatccttgaaagccctggacg
gcgtacttccagatggagtatcggatgcttccagaaccagcgcagatgacaaatggttgcctttgtatct
acttggcttatacagagtgggcagaacacaaatgcctgaatacagaaaaaagctcatggatgggctgaca
aatcaatgcaaaatgatcaatgaacagtttgaacctcttgtgccagaaggtcgtgacatttttgatgtgt
ggggaaatgacagtaattacacaaaaattgtcgctgcagtggacatgttcttccacatgttcaaaaaaca
tgaatgtgcctcgttcagatacggaactattgtttccagattcaaagattgtgctgcattggcaacattt
ggacacctctgcaaaataaccggaatgtctacagaagatgtaacgacctggatcttgaaccgagaagttg
cagatgaaatggtccaaatgatgcttccaggccaagaaattgacaaggccgattcatacatgccttattt
gatcgactttggattgtcttctaagtctccatattcttccgtcaaaaaccctgccttccacttctggggg
caattgacagctcttctgctcagatccaccagagcaaggaatgcccgacagcctgatgacattgagtata
catctcttactacagcaggtttgttgtacgcttatgcagtaggatcctctgccgacttggcacaacagtt
ttgtgttggagataacaaatacactccagatgatagtaccggaggattgacgactaatgcaccgccacaa
ggcagagatgtggtcgaatggctcggatggtttgaagatcaaaacagaaaaccgactcctgatatgatgc
agtatgcgaaaagagcagtcatgtcactgcaaggcctaagagagaagacaattggcaagtatgctaagtc
agaatttgacaaatgagcggccgcactcctcaggtgcaggctgcctatcagaaggtggtggctggtgtgg
ccaatgccctggctcacaaataccactgagatctttttccctctgccaaaaattatggggacatcatgaa
gccccttgagcatctgacttctggctaataaaggaaatttattttcattgcaatagtgtgttggaatttt
ttgtgtctctcactcggaaggacatatgggagggcaaatcatttaaaacatcagaatgagtatttggttt
agagtttggcaacatatgcccatatgctggctgccatgaacaaaggttggctataaagaggtcatcagta
tatgaaacagccccctgctgtccattccttattccatagaaaagccttgacttgaggttagatttttttt
atattttgttttgtgttatttttttctttaacatccctaaaattttccttacatgttttactagccagat
ttttcctcctctcctgactactcccagtcatagctgtccctcttctcttatggagatccctcgacctgca
gcccaagcttggcgtaatcatggtcatagctgtttcctgtgtgaaattgttatccgctcacaattccaca
caacatacgagccggaagcataaagtgtaaagcctggggtgcctaatgagtgagctaactcacattaatt
gcgttgcgctcactgcccgctttccagtcgggaaacctgtcgtgccagcggatccgcatctcaattagtc
agcaaccatagtcccgcccctaactccgcccatcccgcccctaactccgcccagttccgcccattctccg
ccccatggctgactaattttttttatttatgcagaggccgaggccgcctcggcctctgagctattccaga
agtagtgaggaggcttttttggaggcctaggcttttgcaaaaagctaacttgtttattgcagcttataat
ggttacaaataaagcaatagcatcacaaatttcacaaataaagcatttttttcactgcattctagttgtg
gtttgtccaaactcatcaatgtatcttatcatgtctggatccgctgcattaatgaatcggccaacgcgcg
gggagaggcggtttgcgtattgggcgctcttccgcttcctcgctcactgactcgctgcgctcggtcgttc
ggctgcggcgagcggtatcagctcactcaaaggcggtaatacggttatccacagaatcaggggataacgc
aggaaagaacatgtgagcaaaaggccagcaaaaggccaggaaccgtaaaaaggccgcgttgctggcgttt
ttccataggctccgcccccctgacgagcatcacaaaaatcgacgctcaagtcagaggtggcgaaacccga
caggactataaagataccaggcgtttccccctggaagctccctcgtgcgctctcctgttccgaccctgcc
gcttaccggatacctgtccgcctttctcccttcgggaagcgtggcgctttctcatagctcacgctgtagg
tatctcagttcggtgtaggtcgttcgctccaagctgggctgtgtgcacgaaccccccgttcagcccgacc
gctgcgccttatccggtaactatcgtcttgagtccaacccggtaagacacgacttatcgccactggcagc
agccactggtaacaggattagcagagcgaggtatgtaggcggtgctacagagttcttgaagtggtggcct
aactacggctacactagaagaacagtatttggtatctgcgctctgctgaagccagttaccttcggaaaaa
gagttggtagctcttgatccggcaaacaaaccaccgctggtagcggtggtttttttgtttgcaagcagca
gattacgcgcagaaaaaaaggatctcaagaagatcctttgatcttttctacggggtctgacgctcagtgg
aacgaaaactcacgttaagggattttggtcatgagattatcaaaaaggatcttcacctagatccttttaa
attaaaaatgaagttttaaatcaatctaaagtatatatgagtaaacttggtctgacagttaccaatgctt
aatcagtgaggcacctatctcagcgatctgtctatttcgttcatccatagttgcctgactccccgtcgtg
tagataactacgatacgggagggcttaccatctggccccagtgctgcaatgataccgcgagacccacgct
caccggctccagatttatcagcaataaaccagccagccggaagggccgagcgcagaagtggtcctgcaac
tttatccgcctccatccagtctattaattgttgccgggaagctagagtaagtagttcgccagttaatagt
ttgcgcaacgttgttgccattgctacaggcatcgtggtgtcacgctcgtcgtttggtatggcttcattca
gctccggttcccaacgatcaaggcgagttacatgatcccccatgttgtgcaaaaaagcggttagctcctt
cggtcctccgatcgttgtcagaagtaagttggccgcagtgttatcactcatggttatggcagcactgcat
aattctcttactgtcatgccatccgtaagatgcttttctgtgactggtgagtactcaaccaagtcattct
gagaatagtgtatgcggcgaccgagttgctcttgcccggcgtcaatacgggataataccgcgccacatag
cagaactttaaaagtgctcatcattggaaaacgttcttcggggcgaaaactctcaaggatcttaccgctg
ttgagatccagttcgatgtaacccactcgtgcacccaactgatcttcagcatcttttactttcaccagcg
tttctgggtgagcaaaaacaggaaggcaaaatgccgcaaaaaagggaataagggcgacacggaaatgttg
aatactcatactcttcctttttcaatattattgaagcatttatcagggttattgtctcatgagcggatac
atatttgaatgtatttagaaaaataaacaaataggggttccgcgcacatttccccgaaaagtgccacctg
</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>5598</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5598</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtcgacattgattattgactagttattaatagtaatcaattacggggt
cattagttcatagcccatatatggagttccgcgttacataacttacggtaaatggcccgcctggctgacc
gcccaacgacccccgcccattgacgtcaataatgacgtatgttcccatagtaacgccaatagggactttc
cattgacgtcaatgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgc
caagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagtacatgacctt
atgggactttcctacttggcagtacatctacgtattagtcatcgctattaccatggtcgaggtgagcccc
acgttctgcttcactctccccatctcccccccctccccacccccaattttgtatttatttattttttaat
tattttgtgcagcgatgggggcggggggggggggggcgcgcgccaggcggggcggggcggggcgaggggc
ggggcggggcgaggcggagaggtgcggcggcagccaatcagagcggcgcgctccgaaagtttccttttat
ggcgaggcggcggcggcggcggccctataaaaagcgaagcgcgcggcgggcgggagtcgctgcgttgcct
tcgccccgtgccccgctccgcgccgcctcgcgccgcccgccccggctctgactgaccgcgttactcccac
aggtgagcgggcgggacggcccttctcctccgggctgtaattagcgcttggtttaatgacggctcgtttc
ttttctgtggctgcgtgaaagccttaaagggctccgggagggccctttgtgcgggggggagcggctcggg
gggtgcgtgcgtgtgtgtgtgcgtggggagcgccgcgtgcggcccgcgctgcccggcggctgtgagcgct
gcgggcgcggcgcggggctttgtgcgctccgcgtgtgcgcgaggggagcgcggccgggggcggtgccccg
cggtgcgggggggctgcgaggggaacaaaggctgcgtgcggggtgtgtgcgtgggggggtgagcaggggg
tgtgggcgcggcggtcgggctgtaacccccccctgcacccccctccccgagttgctgagcacggcccggc
ttcgggtgcggggctccgtgcggggcgtggcgcggggctcgccgtgccgggcggggggtggcggcaggtg
ggggtgccgggcggggcggggccgcctcgggccggggagggctcgggggaggggcgcggcggccccggag
cgccggcggctgtcgaggcgcggcgagccgcagccattgccttttatggtaatcgtgcgagagggcgcag
ggacttcctttgtcccaaatctgtgcggagccgaaatctgggaggcgccgccgcaccccctctagcgggc
gcggggcgaagcggtgcggcgccggcaggaaggaaatgggcggggagggccttcgtgcgtcgccgcgccg
ccgtccccttctccctctccagcctcggggctgtccgcggggggacggctgccttcgggggggacggggc
agggcggggttcggcttctggcgtgtgaccggcggctctagagcctctgctaaccatgttcatgccttct
tctttttcctacagctcctgggcaacgtgctggttattgtgctgtctcatcattttggcaaagaattcgc
cgccaccatggataatctcacaaaagttcgtgagtatctcaagtcctattctcgtctggatcaggcggta
ggagagatagatgagatcgaagcacaacgagctgaaaagtccaattatgagttgttccaagaggatggag
tggaagagcatactaagccctcttattttcaggcagcagatgattctgacacagaatctgaaccagaaat
tgaagacaatcaaggtttgtatgcacaggatccagaagctgagcaagttgaaggctttatacaggggcct
ttagatgactatgcagatgaggaagtggatgttgtatttacttcggactggaaacagcctgagcttgaat
ctgacgagcatggaaagaccttacggttgacatcgccagagggtttaagtggagagcagaaatcccagtg
gctttcgacgattaaagcagtcgtgcaaagtgccaaatactggaatctggcagagtgcacatttgaagca
tcgggagaaggggtcattatgaaggagcgccagataactccggatgtatataaggtcactccagtgatga
acacacatccgtcccaatcagaagcagtatcagatgtttggtctctctcaaagacatccatgactttcca
acccaagaaagcaagtcttcagcctctcaccatatccttggatgaattgttctcatctagaggagagttc
atctctgtcggaggtgacggacgaatgtctcataaagaggccatcctgctcggcctgagatacaaaaagt
tgtacaatcaggcgagagtcaaatattctctgtaggcggccgcactcctcaggtgcaggctgcctatcag
aaggtggtggctggtgtggccaatgccctggctcacaaataccactgagatctttttccctctgccaaaa
attatggggacatcatgaagccccttgagcatctgacttctggctaataaaggaaatttattttcattgc
aatagtgtgttggaattttttgtgtctctcactcggaaggacatatgggagggcaaatcatttaaaacat
cagaatgagtatttggtttagagtttggcaacatatgcccatatgctggctgccatgaacaaaggttggc
tataaagaggtcatcagtatatgaaacagccccctgctgtccattccttattccatagaaaagccttgac
ttgaggttagattttttttatattttgttttgtgttatttttttctttaacatccctaaaattttcctta
catgttttactagccagatttttcctcctctcctgactactcccagtcatagctgtccctcttctcttat
ggagatccctcgacctgcagcccaagcttggcgtaatcatggtcatagctgtttcctgtgtgaaattgtt
atccgctcacaattccacacaacatacgagccggaagcataaagtgtaaagcctggggtgcctaatgagt
gagctaactcacattaattgcgttgcgctcactgcccgctttccagtcgggaaacctgtcgtgccagcgg
atccgcatctcaattagtcagcaaccatagtcccgcccctaactccgcccatcccgcccctaactccgcc
cagttccgcccattctccgccccatggctgactaattttttttatttatgcagaggccgaggccgcctcg
gcctctgagctattccagaagtagtgaggaggcttttttggaggcctaggcttttgcaaaaagctaactt
gtttattgcagcttataatggttacaaataaagcaatagcatcacaaatttcacaaataaagcatttttt
tcactgcattctagttgtggtttgtccaaactcatcaatgtatcttatcatgtctggatccgctgcatta
atgaatcggccaacgcgcggggagaggcggtttgcgtattgggcgctcttccgcttcctcgctcactgac
tcgctgcgctcggtcgttcggctgcggcgagcggtatcagctcactcaaaggcggtaatacggttatcca
cagaatcaggggataacgcaggaaagaacatgtgagcaaaaggccagcaaaaggccaggaaccgtaaaaa
ggccgcgttgctggcgtttttccataggctccgcccccctgacgagcatcacaaaaatcgacgctcaagt
cagaggtggcgaaacccgacaggactataaagataccaggcgtttccccctggaagctccctcgtgcgct
ctcctgttccgaccctgccgcttaccggatacctgtccgcctttctcccttcgggaagcgtggcgctttc
tcatagctcacgctgtaggtatctcagttcggtgtaggtcgttcgctccaagctgggctgtgtgcacgaa
ccccccgttcagcccgaccgctgcgccttatccggtaactatcgtcttgagtccaacccggtaagacacg
acttatcgccactggcagcagccactggtaacaggattagcagagcgaggtatgtaggcggtgctacaga
gttcttgaagtggtggcctaactacggctacactagaagaacagtatttggtatctgcgctctgctgaag
ccagttaccttcggaaaaagagttggtagctcttgatccggcaaacaaaccaccgctggtagcggtggtt
tttttgtttgcaagcagcagattacgcgcagaaaaaaaggatctcaagaagatcctttgatcttttctac
ggggtctgacgctcagtggaacgaaaactcacgttaagggattttggtcatgagattatcaaaaaggatc
ttcacctagatccttttaaattaaaaatgaagttttaaatcaatctaaagtatatatgagtaaacttggt
ctgacagttaccaatgcttaatcagtgaggcacctatctcagcgatctgtctatttcgttcatccatagt
tgcctgactccccgtcgtgtagataactacgatacgggagggcttaccatctggccccagtgctgcaatg
ataccgcgagacccacgctcaccggctccagatttatcagcaataaaccagccagccggaagggccgagc
gcagaagtggtcctgcaactttatccgcctccatccagtctattaattgttgccgggaagctagagtaag
tagttcgccagttaatagtttgcgcaacgttgttgccattgctacaggcatcgtggtgtcacgctcgtcg
tttggtatggcttcattcagctccggttcccaacgatcaaggcgagttacatgatcccccatgttgtgca
aaaaagcggttagctccttcggtcctccgatcgttgtcagaagtaagttggccgcagtgttatcactcat
ggttatggcagcactgcataattctcttactgtcatgccatccgtaagatgcttttctgtgactggtgag
tactcaaccaagtcattctgagaatagtgtatgcggcgaccgagttgctcttgcccggcgtcaatacggg
ataataccgcgccacatagcagaactttaaaagtgctcatcattggaaaacgttcttcggggcgaaaact
ctcaaggatcttaccgctgttgagatccagttcgatgtaacccactcgtgcacccaactgatcttcagca
tcttttactttcaccagcgtttctgggtgagcaaaaacaggaaggcaaaatgccgcaaaaaagggaataa
gggcgacacggaaatgttgaatactcatactcttcctttttcaatattattgaagcatttatcagggtta
ttgtctcatgagcggatacatatttgaatgtatttagaaaaataaacaaataggggttccgcgcacattt
ccccgaaaagtgccacctg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>2025</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..2025</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atggaaacagatacactcctgctctgggtgctgctgctgtgggtccctg
gctccaccggcagaaacctgcctgtggccacccccgatcccggcatgttcccttgtctgcaccacagcca
gaatctgctgagagccgtgagcaacatgctgcaaaaggccagacagaccctggagttctatccttgcacc
tctgaagagatcgaccacgaggacatcaccaaggacaagacatctacagtggaagcctgtctgccactgg
aactgacaaagaacgagagctgtctgaacagccgggaaaccagctttatcaccaacggctcttgcctcgc
ctctcggaagactagcttcatgatggccctgtgtctgagcagcatctacgaggacctgaaaatgtaccag
gtggagttcaagaccatgaacgctaaactgctgatggaccctaaaagacaaatcttcctggatcagaaca
tgctggccgtgattgacgagctgatgcaggctctgaatttcaacagcgagacagtgccccaaaaaagcag
tctggaagaacctgatttctacaagaccaagatcaagctgtgcatcctgctgcacgccttcagaatccgg
gccgttacaatcgacagagttatgagctacctgaacgcctccggcggcggcggatctggcggcggaggct
ctggcggaggtggaagcatctgggaactgaaaaaggatgtgtatgtggtggaactggactggtatcctga
cgcccctggcgagatggtggtgctgacatgcgacacccccgaggaagatggcatcacatggaccctcgac
cagagctctgaggtgctgggctctggaaagaccctgaccatccaggtgaaggagttcggcgatgccggcc
agtacacatgccacaagggcggcgaagtgctgagccacagcctgctgttgctgcataagaaagaggacgg
catctggagcaccgacatcctgaaggaccagaaagagcctaagaacaagacttttctgcggtgcgaggcc
aagaactacagcggaagattcacctgttggtggctgacgaccattagcaccgacctgaccttttctgtga
agtctagcaggggcagcagcgacccccagggcgtgacctgcggcgccgctaccctgtccgccgagagagt
gcggggcgacaacaaggaatacgagtacagtgtggagtgccaggaggatagcgcctgccctgctgctgag
gaaagcctgcccatcgaggtgatggtggacgccgtgcacaagctgaagtacgagaactacacctcatctt
tcttcatcagagatatcatcaagcctgacccacctaagaacctgcaactgaagcccctgaaaaattcaag
acaggtggaagtgagctgggagtatcctgatacctggagcacccctcactcctacttctcactgaccttc
tgcgtgcaggttcagggcaagtccaagagagagaagaaggacagagtctttacagacaaaaccagcgcca
ccgtgatctgccggaagaatgccagcatcagcgtgcgggcccaggaccgctactactccagctcttggtc
cgagtgggccagcgtcccttgcagcggaggaggtggttctggaggtggtggttcggcccctgctagatcc
cctagccccagcacccagccttgggagcacgtgaacgccatccaggaggccagacggctgcttaatctga
gcagagacaccgccgctgaaatgaacgagacagtggaagtgatcagcgagatgttcgacctgcaagagcc
tacctgcctgcaaaccagactggaactgtacaagcagggcctgcggggatctctgaccaagctgaaaggc
cctctgaccatgatggccagccactacaagcaacactgtcctccaacacctgagacaagctgcgccaccc
agatcatcaccttcgagagctttaaggaaaacctgaaggacttcctgctcgtgatccccttcgactgctg
ggagcccgtgcaggag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>2061</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..2061</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atggaaacagatacactcctgctctgggtgctgctgctgtgggtccctg
gctccaccggcagaaacctgcctgtggccacccccgatcccggcatgttcccttgtctgcaccacagcca
gaatctgctgagagccgtgagcaacatgctgcaaaaggccagacagaccctggagttctatccttgcacc
tctgaagagatcgaccacgaggacatcaccaaggacaagacatctacagtggaagcctgtctgccactgg
aactgacaaagaacgagagctgtctgaacagccgggaaaccagctttatcaccaacggctcttgcctcgc
ctctcggaagactagcttcatgatggccctgtgtctgagcagcatctacgaggacctgaaaatgtaccag
gtggagttcaagaccatgaacgctaaactgctgatggaccctaaaagacaaatcttcctggatcagaaca
tgctggccgtgattgacgagctgatgcaggctctgaatttcaacagcgagacagtgccccaaaaaagcag
tctggaagaacctgatttctacaagaccaagatcaagctgtgcatcctgctgcacgccttcagaatccgg
gccgttacaatcgacagagttatgagctacctgaacgcctccggcggcggcggatctggcggcggaggct
ctggcggaggtggaagcatctgggaactgaaaaaggatgtgtatgtggtggaactggactggtatcctga
cgcccctggcgagatggtggtgctgacatgcgacacccccgaggaagatggcatcacatggaccctcgac
cagagctctgaggtgctgggctctggaaagaccctgaccatccaggtgaaggagttcggcgatgccggcc
agtacacatgccacaagggcggcgaagtgctgagccacagcctgctgttgctgcataagaaagaggacgg
catctggagcaccgacatcctgaaggaccagaaagagcctaagaacaagacttttctgcggtgcgaggcc
aagaactacagcggaagattcacctgttggtggctgacgaccattagcaccgacctgaccttttctgtga
agtctagcaggggcagcagcgacccccagggcgtgacctgcggcgccgctaccctgtccgccgagagagt
gcggggcgacaacaaggaatacgagtacagtgtggagtgccaggaggatagcgcctgccctgctgctgag
gaaagcctgcccatcgaggtgatggtggacgccgtgcacaagctgaagtacgagaactacacctcatctt
tcttcatcagagatatcatcaagcctgacccacctaagaacctgcaactgaagcccctgaaaaattcaag
acaggtggaagtgagctgggagtatcctgatacctggagcacccctcactcctacttctcactgaccttc
tgcgtgcaggttcagggcaagtccaagagagagaagaaggacagagtctttacagacaaaaccagcgcca
ccgtgatctgccggaagaatgccagcatcagcgtgcgggcccaggaccgctactactccagctcttggtc
cgagtgggccagcgtcccttgcagcggaggcggaggcagtggtggcggagggtccggtggcggaggtagt
ggaggaggaggttcaggctcagcccctgctagatcccctagccccagcacccagccttgggagcacgtga
acgccatccaggaggccagacggctgcttaatctgagcagagacaccgccgctgaaatgaacgagacagt
ggaagtgatcagcgagatgttcgacctgcaagagcctacctgcctgcaaaccagactggaactgtacaag
cagggcctgcggggatctctgaccaagctgaaaggccctctgaccatgatggccagccactacaagcaac
actgtcctccaacacctgagacaagctgcgccacccagatcatcaccttcgagagctttaaggaaaacct
gaaggacttcctgctcgtgatccccttcgactgctgggagcccgtgcaggag</INSDSeq_sequence
>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
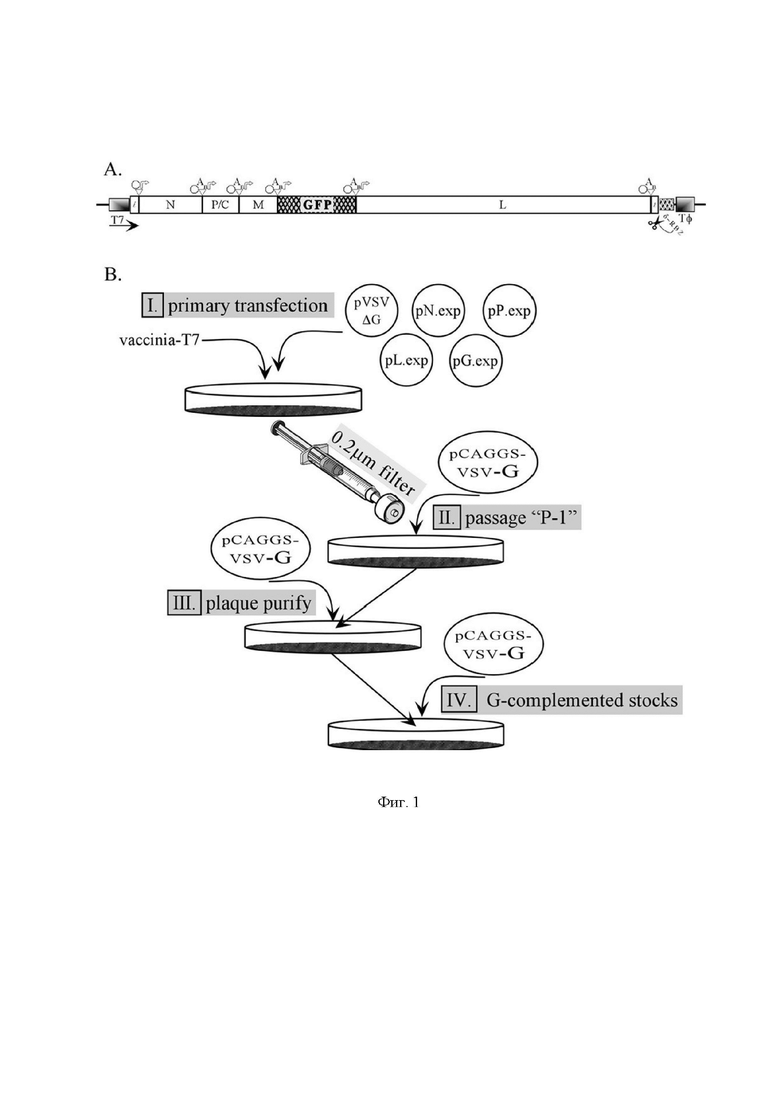
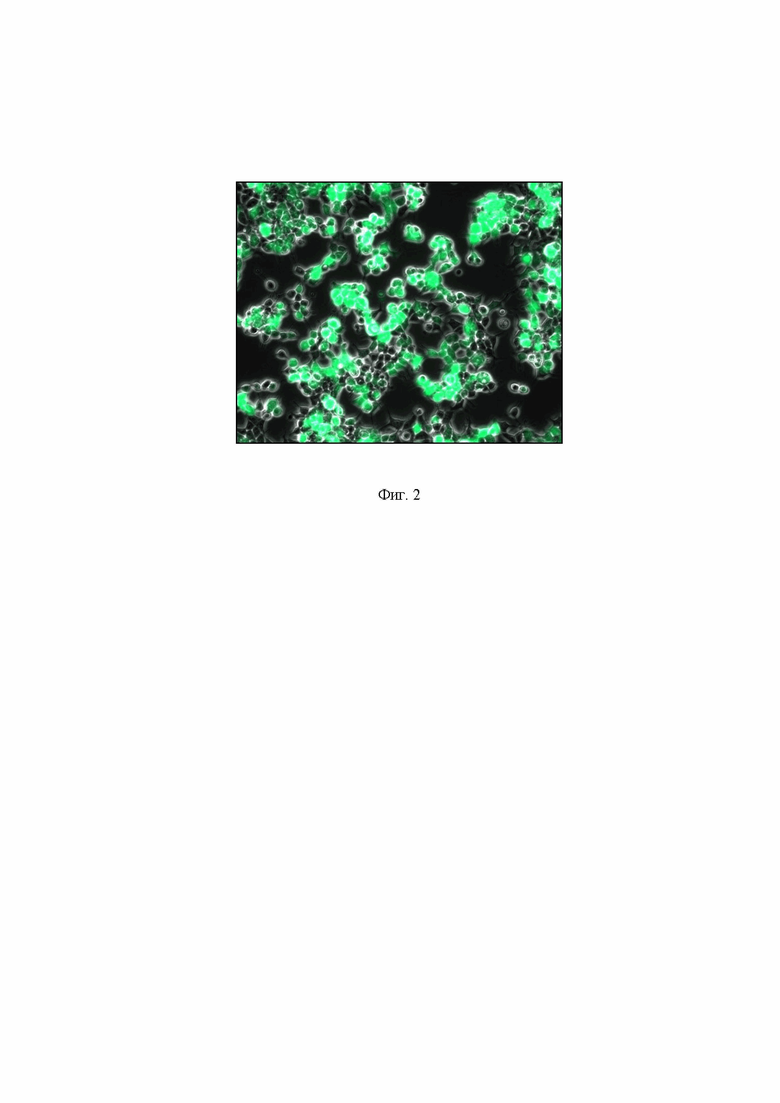
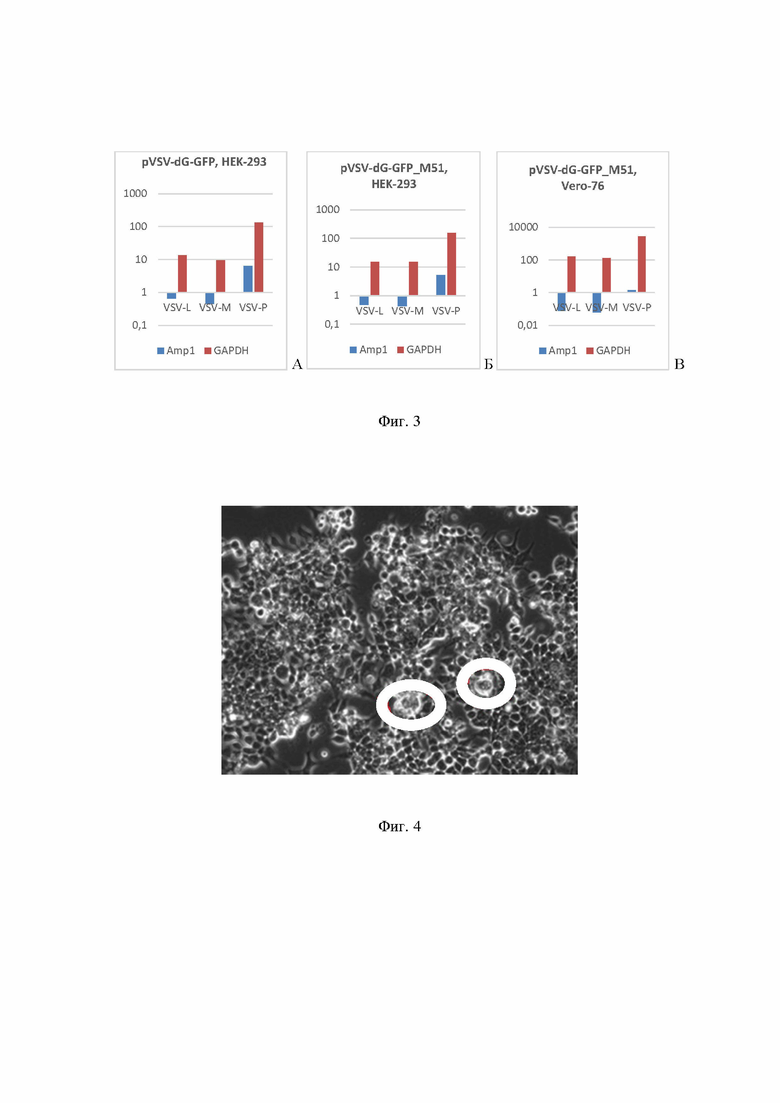

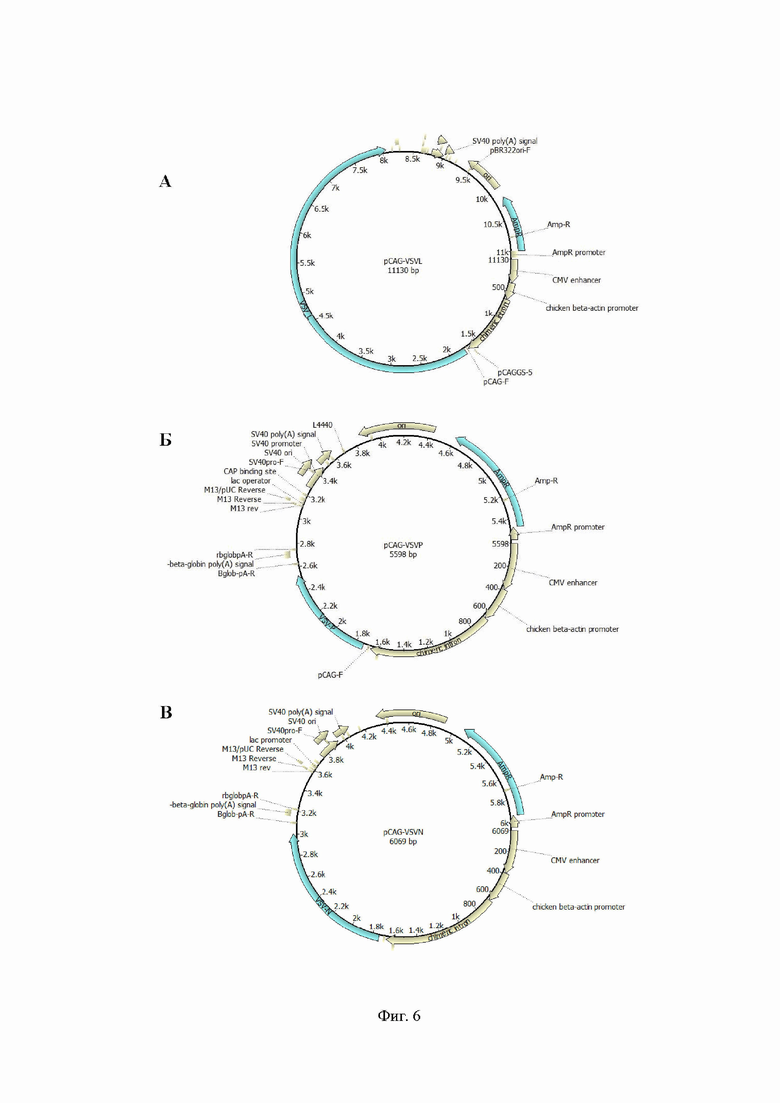
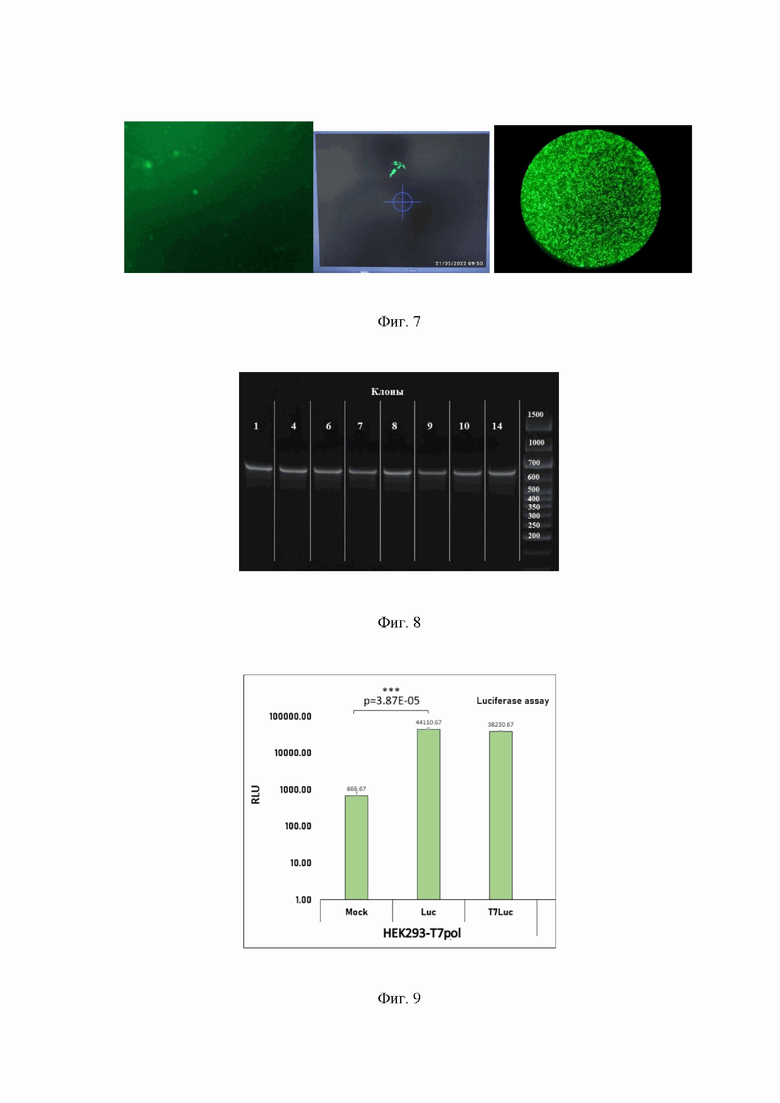
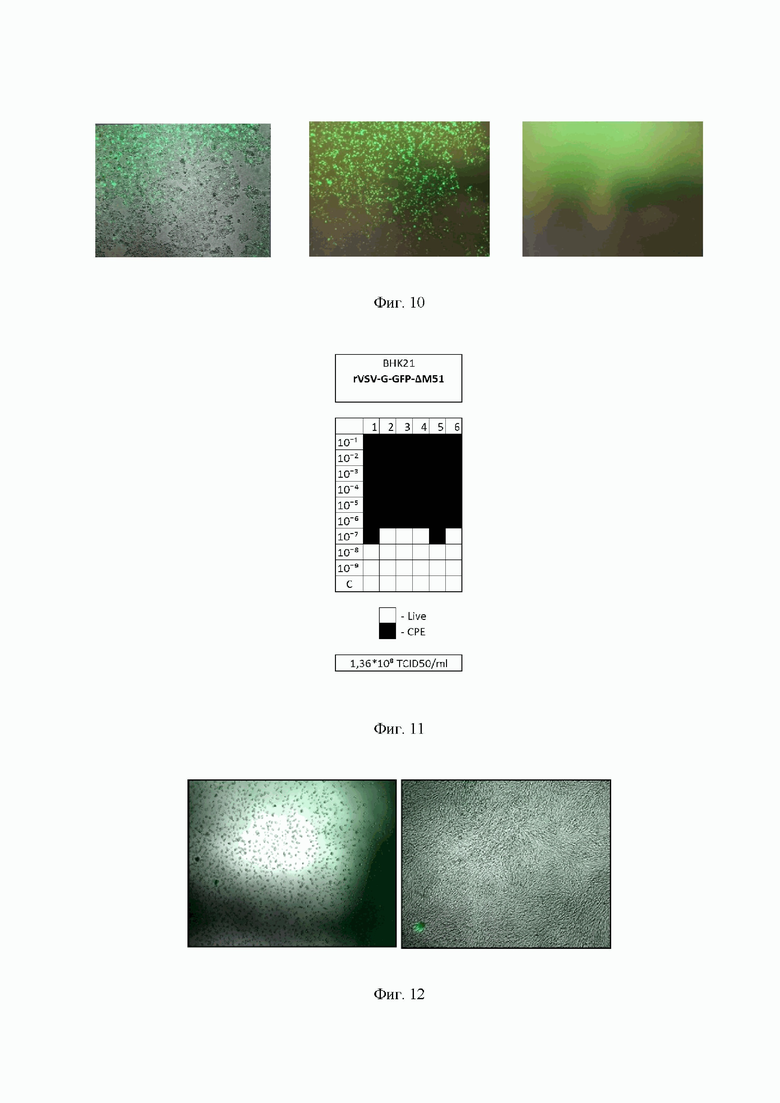
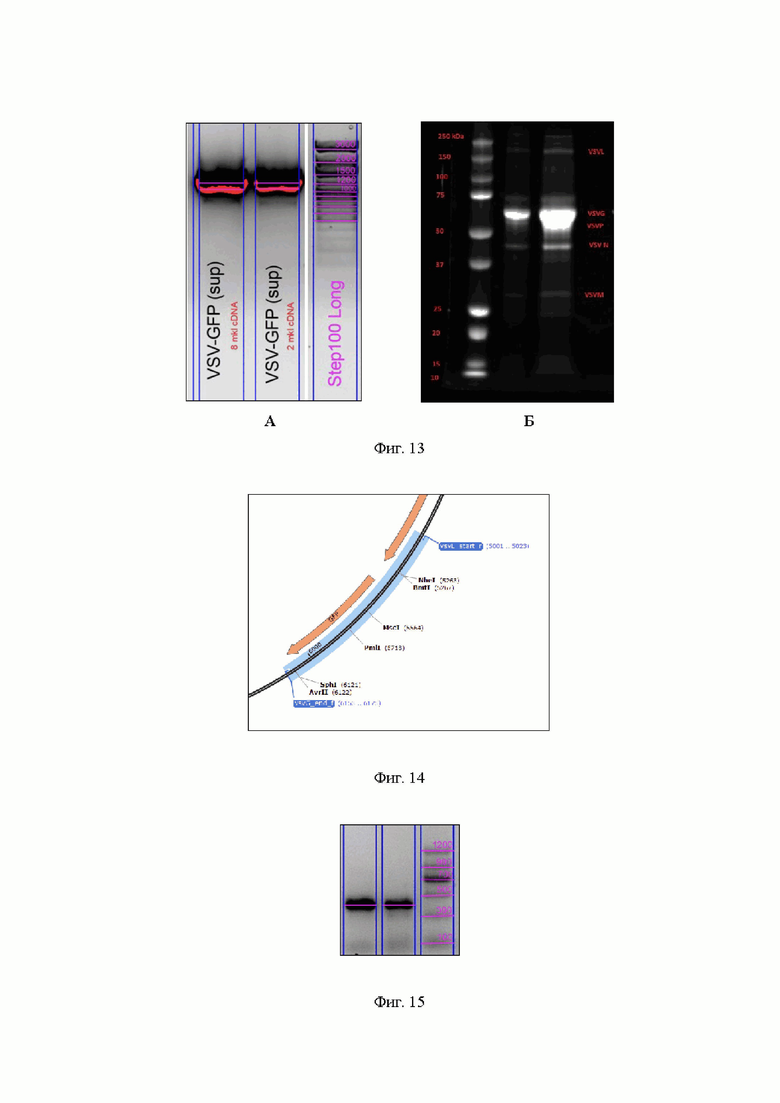
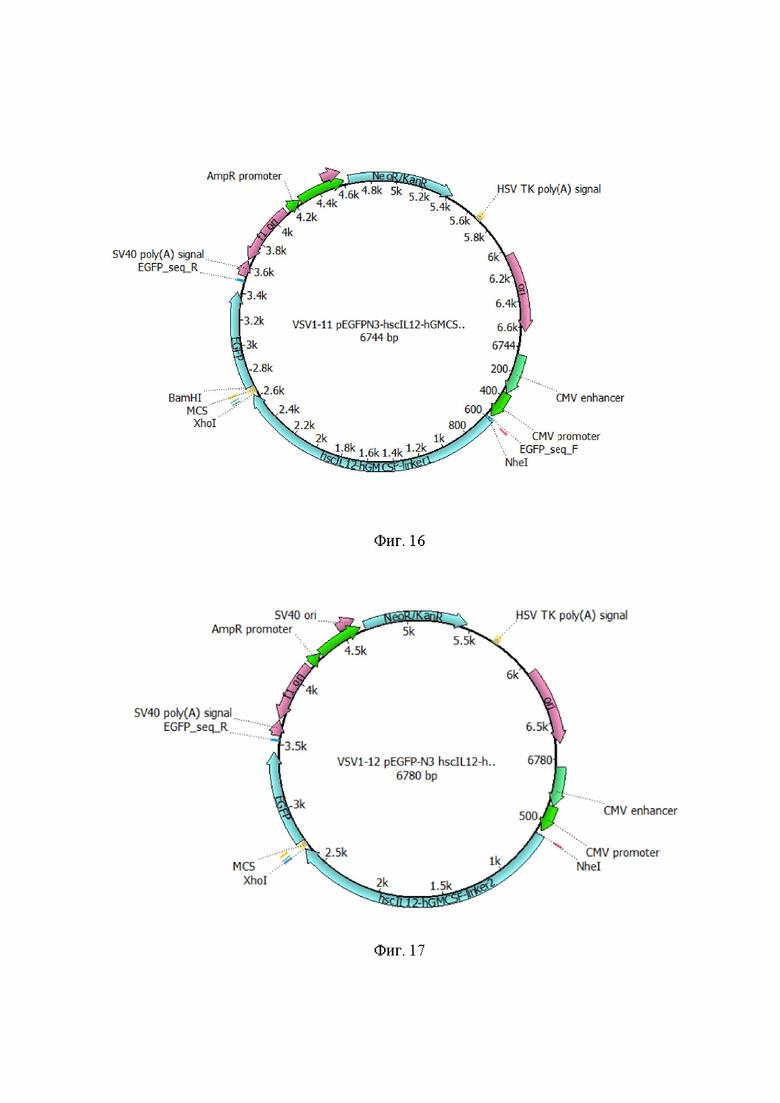
Изобретение относится к биотехнологии. Описан способ получения рекомбинантного вируса везикулярного стоматита. Клетки, стабильно экспрессирующие ДНК-зависимую РНК-полимеразу бактериофага Т7, трансфицируют с использованием смеси плазмидных экспрессионных векторов и полиэтиленимина в весовом соотношении ДНК: полиэтиленимин 5:1. При этом упомянутая смесь содержит четыре плазмидных экспрессионных вектора pCAG-VSVN, pCAG-VSVP, pCAG-VSVL и pCAG-VSVG, каждый из которых кодирует один из белков вируса везикулярного стоматита N, P, L, G соответственно под контролем промотора CAG, и плазмидный экспрессионный вектор, кодирующий антигеномно-смысловую РНК генома вируса везикулярного стоматита под контролем промотора T7, в соотношении 3:5:1:4:8 или 3:5:1:8:10. Культивируют, собирают культуральную жидкость, этой жидкостью трансдуцируют другие клетки, культивируют их, собирают кондиционированную культуральную жидкость. 17 з.п. ф-лы, 17 ил., 9 пр.
1. Способ получения вирусных частиц рекомбинантного вируса везикулярного стоматита, характеризующийся тем, что
клетки, стабильно экспрессирующие ДНК-зависимую РНК-полимеразу бактериофага Т7,
трансфицируют с использованием смеси плазмидных экспрессионных векторов и полиэтиленимина в весовом соотношении ДНК: полиэтиленимин 5:1, при этом упомянутая смесь содержит четыре плазмидных экспрессионных вектора pCAG-VSVN, pCAG-VSVP, pCAG-VSVL и pCAG-VSVG, каждый из которых кодирует один из белков вируса везикулярного стоматита N, P, L, G соответственно под контролем промотора CAG, и плазмидный экспрессионный вектор, кодирующий антигеномно-смысловую РНК генома вируса везикулярного стоматита под контролем промотора T7, в соотношении 3:5:1:4:8 или 3:5:1:8:10,
культивируют, собирают культуральную жидкость, этой жидкостью трансдуцируют другие клетки, культивируют их, собирают кондиционированную культуральную жидкость.
2. Способ по п. 1, отличающийся тем, что полученной кондиционированной культуральной жидкостью дополнительно трансдуцируют клетки, культивируют, собирают кондиционированную культуральную жидкость.
3. Способ по п. 2, отличающийся тем, что дополнительную трансдукцию проводят два раза и более.
4. Способ по любому из пп. 1-3, отличающийся тем, что клетки, стабильно экспрессирующие ДНК-зависимую РНК-полимеразу бактериофага Т7, представляют собой клетки линии HEK293TN, стабильно экспрессирующие ДНК-зависимую РНК-полимеразу бактериофага Т7.
5. Способ по п. 4, отличающийся тем, что клетки линии HEK293TN, стабильно экспрессирующие ДНК-зависимую РНК-полимеразу бактериофага Т7, получены путём трансдукции клеток линии HEK293TN ретровирусом, несущим ген ДНК-зависимой РНК-полимеразы бактериофага Т7.
6. Способ по любому из пп. 1-3, отличающийся тем, что клетки, стабильно экспрессирующие ДНК-зависимую РНК-полимеразу бактериофага Т7, представляют собой клетки линии BHK21 T7, стабильно экспрессирующие ДНК-зависимую РНК-полимеразу бактериофага Т7.
7. Способ по п. 6, отличающийся тем, что клетки линии BHK21 T7, стабильно экспрессирующие ДНК-зависимую РНК-полимеразу бактериофага Т7, получены путём трансдукции клеток линии BHK21 ретровирусом, несущим ген ДНК-зависимой РНК-полимеразы бактериофага Т7.
8. Способ по любому из пп. 1-7, отличающийся тем, что смесь плазмидных экспрессионных векторов дополнительно содержит плазмидный экспрессионный вектор, кодирующий ДНК-зависимую РНК-полимеразу бактериофага Т7, при этом векторы, кодирующие белки вируса везикулярного стоматита N, P, L, G, вектор, кодирующий ДНК-зависимую РНК-полимеразу бактериофага Т7, и вектор, кодирующий антигеномно-смысловую РНК генома вируса везикулярного стоматита, содержатся в упомянутой смеси в соотношении 3:5:1:4:5:8 или 3:5:1:8:10:10.
9. Способ по любому из пп. 1-8, характеризующийся тем, что плазмидный экспрессионный вектор, кодирующий антигеномно-смысловую РНК генома вируса везикулярного стоматита, содержит гетерологичный ген, кодирующий белок интереса.
10. Способ по п. 9, характеризующийся тем, что белок интереса представляет собой зеленый флюоресцентный белок, или химерный белок.
11. Способ по любому из пп. 1-10, отличающийся тем, что плазмидный экспрессионный вектор, кодирующий белок вируса везикулярного стоматита N, представляет собой вектор, содержащий в соответствии с физической и генетической картой, представленной на Фиг. 6В, следующие элементы:
энхансер цитомегаловирусного промотора CMV enhancer;
промотор бета-актина цыплёнка Chicken β-actin promoter;
химерный интрон, увеличивающий экспрессию трансгена Chimeric intron;
последовательность гена белка VSV-N нуклеокапсида вируса везикулярного стоматита VSV-N;
сигнал полиаденилирования β-globin poly(A) signal;
Lac-оператор Lac operator;
Lac-промотор Lac promoter;
промотор SV40 SV40 promoter;
участок начала репликации SV40 ori;
сигнал полиаденилирования SV40 Poly(A)signal;
участок начала репликации ori;
ген устойчивости к ампициллину AmpR;
промотор гена устойчивости к ампициллину AmpR promoter.
12. Способ по п. 11, отличающийся тем, что плазмидный экспрессионный вектор, кодирующий белок вируса везикулярного стоматита N, содержит последовательность SEQ Id No.: 5.
13. Способ по любому из пп. 1-10, отличающийся тем, что плазмидный экспрессионный вектор, кодирующий белок вируса везикулярного стоматита P, представляет собой вектор, содержащий в соответствии с физической и генетической картой, представленной на Фиг. 6Б, следующие элементы:
энхансер цитомегаловирусного промотора CMV enhancer;
промотор бета-актина цыплёнка Chicken β-actin promoter;
химерный интрон, увеличивающий экспрессию трансгена Chimeric intron;
последовательность гена белка VSV-P фосфопротеина вируса везикулярного стоматита VSV-P;
сигнал полиаденилирования β-globin poly(A) signal;
Lac-оператор Lac operator;
Lac-промотор Lac promoter;
промотор SV40 SV40 promoter;
участок начала репликации SV40 ori;
сигнал полиаденилирования SV40 Poly(A)signal;
участок начала репликации ori;
ген устойчивости к ампициллину AmpR;
промотор гена устойчивости к ампициллину AmpR promoter.
14. Способ по п. 13, отличающийся тем, что плазмидный экспрессионный вектор, кодирующий белок вируса везикулярного стоматита P, содержит последовательность SEQ Id No.: 6.
15. Способ по любому из пп. 1-10, отличающийся тем, что плазмидный экспрессионный вектор, кодирующий белок вируса везикулярного стоматита L, представляет собой вектор, содержащий в соответствии с физической и генетической картой, представленной на Фиг. 6А, следующие элементы:
энхансер цитомегаловирусного промотора CMV enhancer;
промотор бета-актина цыплёнка Chicken β-actin promoter;
химерный интрон, увеличивающий экспрессию трансгена Chimeric intron;
последовательность гена белка VSV-L большой субъединицы полимеразы вируса везикулярного стоматита VSV-L;
сигнал полиаденилирования β-globin poly(A) signal;
Lac-оператор Lac operator;
Lac-промотор Lac promoter;
промотор SV40 SV40 promoter;
участок начала репликации SV40 ori;
сигнал полиаденилирования SV40 Poly(A)signal;
участок начала репликации ori;
ген устойчивости к ампициллину AmpR;
промотор гена устойчивости к ампициллину AmpR promoter.
16. Способ по п. 15, отличающийся тем, что плазмидный экспрессионный вектор, кодирующий белок вируса везикулярного стоматита L, содержит последовательность SEQ Id No.: 4.
17. Способ по любому из пп. 1-16, отличающийся тем, что трансфекцию проводят в лунках 12-луночного планшета, при этом общее количество плазмидной ДНК при расчете на лунку составляет 10,5 мкг при соотношении векторов, кодирующих белки N, P, L, G и антигеномно-смысловую РНК генома вируса везикулярного стоматита 3:5:1:4:8.
18. Способ по любому из пп. 1-16, отличающийся тем, что трансфекцию проводят в лунках 12-луночного планшета, при этом общее количество плазмидной ДНК при расчете на лунку составляет 6,75 мкг при соотношении векторов, кодирующих белки N, P, L, G и антигеномно-смысловую РНК генома вируса везикулярного стоматита 3:5:1:8:10.
| СПОСОБ ПОЛУЧЕНИЯ ПРЕПАРАТИВНЫХ КОЛИЧЕСТВ ВИРУСНЫХ ЧАСТИЦ ФЛОЭМНО-ОРГАНИЧЕННЫХ ВИРУСОВ | 2013 |
|
RU2555534C2 |
| RU 2020101596 A, 27.07.2021 | |||
| ПЛАЗМИДЫ И СПОСОБ ПОЛУЧЕНИЯ ВИРУСНЫХ ЧАСТИЦ | 2016 |
|
RU2708151C2 |
| ВИРУСНЫЕ ЧАСТИЦЫ, СОДЕРЖАЩИЕ ВЕКТОР, ПРОИСХОДЯЩИЙ ИЗ АЛЬФАВИРУСА, И СПОСОБ ПОЛУЧЕНИЯ УКАЗАННОЙ ВИРУСНОЙ ЧАСТИЦЫ | 2004 |
|
RU2398875C2 |
| WO 2014145578 A1, 18.09.2014. | |||
Авторы
Даты
2023-11-21—Публикация
2022-12-27—Подача