ОБЛАСТЬ ТЕХНИКИ, К КОТОРОЙ ОТНОСИТСЯ ИЗОБРЕТЕНИЕ
Настоящее изобретение относится, среди прочего, к вегетативным частям растений, из растения вида Sorghum и/или Zea mays, имеющим общее содержание жирных кислот (TFA), включающее жирные кислоты, этерифицированные в форме триацилглицеринов (TAG), и жирные кислоты в форме липидов, отличных от TAG, где вегетативные части растений имеют намного увеличенные уровни TFA, например, содержание TFA приблизительно 5% (масс./масс. сухой массы). Настоящее изобретение также относится к применению вегетативных частей растений в качестве пищевого продукта и/или для получения пищевого продукта, для употребления животными.
УРОВЕНЬ ТЕХНИКИ ДЛЯ ИЗОБРЕТЕНИЯ
Удовлетворение запросов потребителей в отношении продуктов животноводства, например, мяса, молока и яиц, зависит от доступности регулярных поставок безопасных, экономически эффективных кормов для животных, в частности, высокоэнергетических кормов, таких как корма с высокими уровнями жирных кислот. По мере увеличения запросов потребителей в отношении таких продуктов животноводства, в частности, в развивающихся странах, например, предполагают увеличение общемировой потребности в мясных продуктах на 58% между 1995 и 2020 гг. (FAO Animal Production and Health Proceedings, 2002), необходимо увеличение поставок кормового белка.
Существует необходимость в вегетативных частях растений, в частности, вегетативных частях растений из важных кормовых зерновых культур, таких как сорго и кукуруза, с высоким общим содержанием жирных кислот.
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
Настоящее изобретение относится к растениям и вегетативным части растений, предпочтительно, из вида Sorghum и/или Zea mays, с увеличенным общим содержанием жирных кислот, и к их применениям.
Таким образом, в первом аспекте, настоящее изобретение относится к способу получения пищевого продукта для животного, включающему стадии
(i) сбора вегетативных частей растений из растения вида Sorghum и/или Zea mays, имеющих общее содержание жирных кислот (TFA), включающее жирные кислоты, этерифицированные в форме триацилглицеринов (TAG), и жирные кислоты в форме липидов, отличных от TAG, где вегетативные части растений имеют содержание TFA приблизительно 5% (масс./масс. сухой массы), и одну или несколько из стадий
(ii) смешивания собранных частей растений по меньшей мере с одним другим кормовым ингредиентом,
(iii) упаковки в тюки собранных частей растений,
(iv) переработки собранных частей растений, предпочтительно, посредством рубки, нарезки, сушки, прессования или гранулирования частей растений, в форму, пригодную для употребления животным, и
(v) хранения собранных частей растений в условиях уменьшенного содержания кислорода в течение такого периода времени, что по меньшей мере некоторые из углеводов в частях растений подвергаются ферментации до органических кислот.
В одном варианте осуществления, вегетативные части растений имеют соотношение TAG/TFA (TTQ) между 0,01 и 0,6. В одном варианте осуществления, вегетативные части растений имеют TTQ между 0,01 и 0,55, или между 0,01 и 0,5, или приблизительно 0,1, или приблизительно 0,2 или приблизительно 0,3, или приблизительно 0,4, или приблизительно 0,5. Предпочтительно, TTQ составляет между 0,60 и 0,84, что соответствует соотношению TAG:TFA между 1,5:1 и 5:1, или между 0,84 и 0,95, что соответствует соотношению TAG:TFA между 5:1 и 20:1.
В одном варианте осуществления, вегетативные части растений имеют среднее содержание TFA приблизительно 6% или приблизительно 8%, или приблизительно 9%, или приблизительно 10% (масс./масс. сухой массы).
В одном варианте осуществления, содержание TFA в вегетативных частях растений включает содержание олеиновой кислоты, которое увеличено по меньшей мере на 2% или по меньшей мере на 3% относительно содержания олеиновой кислоты в соответствующей вегетативной части растения дикого типа.
В одном варианте осуществления, содержание TFA в вегетативных частях растений включает содержание пальмитиновой кислоты, которое увеличено по меньшей мере на 2% или по меньшей мере на 3% относительно содержания пальмитиновой кислоты в соответствующей вегетативной части растения дикого типа.
В одном варианте осуществления, содержание TFA в вегетативных частях растений включает содержание α-линолеиновой кислоты (ALA), которое уменьшено по меньшей мере на 2% или по меньшей мере на 3% относительно содержания ALA в соответствующей вегетативной части растения дикого типа.
В одном варианте осуществления используют один или несколько, или все из следующих признаков:
(i) вегетативные части растений представляют собой листья и/или стебли, или их части, имеющие одно или несколько из увеличенного содержания углерода, увеличенного содержания энергии, увеличенного содержания растворимого белка, уменьшенного содержания крахмала, уменьшенного общего содержания пищевых волокон (TDF) и увеличенного содержания азота, каждое в пересчете на массу, по сравнению с соответствующим листом или стеблем дикого типа, или их частями, из растения вида Sorghum и/или Zea mays дикого типа на такой же стадии роста,
(ii) содержание TFA в вегетативных частях растений составляет по меньшей мере приблизительно 6%, по меньшей мере приблизительно 7%, по меньшей мере приблизительно 8%, по меньшей мере приблизительно 9%, по меньшей мере приблизительно на 10%, по меньшей мере приблизительно 11%, по меньшей мере приблизительно 12%, по меньшей мере приблизительно 15%, по меньшей мере приблизительно 20%, по меньшей мере приблизительно на 25%, по меньшей мере приблизительно 30%, по меньшей мере приблизительно 35%, по меньшей мере приблизительно 40%, по меньшей мере приблизительно 45%, по меньшей мере приблизительно на 50%, по меньшей мере приблизительно 55%, по меньшей мере приблизительно 60%, по меньшей мере приблизительно 65%, по меньшей мере приблизительно 70%, между приблизительно 6% и приблизительно 20%, между 8% и 75%, между 10% и 75%, между 11% и 75%, между приблизительно 15% и 75%, между приблизительно 20% и 75%, между приблизительно 30% и 75%, между приблизительно 40% и 75%, между приблизительно 50% и 75%, между приблизительно 60% и 75%, или между приблизительно 25% и 50% (масс./масс. сухой массы) TFA,
(iii) жирные кислоты, этерифицированные в форме TAG, в вегетативных частях растений составляют по меньшей мере приблизительно 1%, по меньшей мере приблизительно 2%, по меньшей мере приблизительно 3%, по меньшей мере приблизительно 4%, по меньшей мере приблизительно 5%, по меньшей мере приблизительно 6%, по меньшей мере приблизительно 7%, по меньшей мере приблизительно 8%, по меньшей мере приблизительно 9%, по меньшей мере приблизительно на 10%, по меньшей мере приблизительно 11%, по меньшей мере приблизительно 12%, по меньшей мере приблизительно 15%, по меньшей мере приблизительно 20%, по меньшей мере приблизительно на 25%, по меньшей мере приблизительно 30%, по меньшей мере приблизительно 35%, по меньшей мере приблизительно 40%, по меньшей мере приблизительно 45%, по меньшей мере приблизительно на 50%, по меньшей мере приблизительно 55%, по меньшей мере приблизительно 60%, по меньшей мере приблизительно 65%, по меньшей мере приблизительно 70%, между приблизительно 6% и приблизительно 20%, между 8% и 75%, между 10% и 75%, между 11% и 75%, между приблизительно 15% и 75%, между приблизительно 20% и 75%, между приблизительно 30% и 75%, между приблизительно 40% и 75%, между приблизительно 50% и 75%, между приблизительно 60% и 75%, или между приблизительно 25% и 50% (масс./масс. сухой массы),
(iv) вегетативные части растений имеют увеличенное содержание полипептида WRI1, увеличенное содержание полипептида DGAT и уменьшенное содержание полипептида SDP1, каждое по сравнению с соответствующей вегетативной частью растения дикого типа,
(v) вегетативные части растений имеют увеличенное содержание полипептида WRI1, увеличенное содержание полипептида DGAT и увеличенное содержание полипептида LEC2, каждое по сравнению с соответствующей вегетативной частью растения дикого типа,
(vi) вегетативные части растений имеют увеличенное содержание полипептида PDAT или DGAT, уменьшенное содержание полипептида TGD, и уменьшенное содержание полипептида SDP1, каждое по сравнению с соответствующей вегетативной частью растения дикого типа, и
(vii) вегетативные части растений имеют уменьшенное содержание TAG-липазы, такой как TAG-липаза SDP1, уменьшенное содержание полипептида TGD, такого как полипептид TGD5, и необязательно, уменьшенное содержание полипептида TST, такого как полипептид TST1, где каждое уменьшение присутствует по сравнению с соответствующей вегетативной частью растения дикого типа.
Во втором аспекте, настоящее изобретение относится к способу получения пищевого продукта для животного, включающему стадии
(i) сбора вегетативных частей растений из растения вида Sorghum и/или Zea mays, имеющих общее содержание жирных кислот включающее жирные кислоты, этерифицированные в форме триацилглицеринов (TAG), и жирные кислоты в форме липидов, отличных от TAG, где вегетативные части растений имеют общее содержание TAG приблизительно 6% (масс./масс. сухой массы) и предпочтительно, имеют соотношение жирных кислот, этерифицированных в форме TAG, к жирным кислотам в форме липидов, отличных от TAG, составляющее между 20:1 и 1,5:1 или между 5:1 и 2:1, и одну или несколько из стадий
(ii) смешивания собранных частей растений по меньшей мере с одним другим кормовым ингредиентом,
(iii) упаковку в тюки собранных частей растений,
(iv) переработку собранных частей растений, предпочтительно, посредством рубки, нарезки, сушки, прессования или гранулирования частей растений, в форму, пригодную для употребления животным, и
(v) хранения собранных частей растений в условиях уменьшенного содержания кислорода в течение такого периода времени, что по меньшей мере некоторые из углеводов в частях растений подвергаются ферментации до органических кислот.
В одном варианте осуществления двух вышеуказанных аспектов, используют один или несколько, или все из следующих признаков:
(i) вегетативные части растений собирают из растения между временем первого цветения растения и первого созревания семян,
(ii) растение вида Sorghum представляет собой растение Sorghum bicolor,
(iii) вегетативные части растений включают листья и/или стебли, или их части,
(iv) вегетативные части растений имеют среднее общее содержание жирных кислот приблизительно 8% или приблизительно 10% (масс./масс. сухой массы),
(v) общее содержание жирных кислот в вегетативных частях растений включает содержание олеиновой кислоты, которое увеличено по меньшей мере на 2% или по меньшей мере на 3% относительно содержания олеиновой кислоты в соответствующей вегетативной части растения дикого типа,
(vi) общее содержание жирных кислот в вегетативных частях растений включает содержание пальмитиновой кислоты, которое увеличено по меньшей мере на 2% или по меньшей мере на 3% относительно содержания пальмитиновой кислоты в соответствующей вегетативной части растения дикого типа,
(vii) общее содержание жирных кислот в вегетативных частях растений включает содержание α-линолеиновой кислоты (ALA), которое уменьшено по меньшей мере на 2% или по меньшей мере на 3% относительно содержания ALA соответствующей вегетативной части растения дикого типа,
(viii) вегетативные части растений имеют увеличенное содержание растворимого белка по сравнению с соответствующей вегетативной частью растения дикого типа,
(ix) вегетативные части растений имеют увеличенное содержание азота по сравнению с соответствующей вегетативной частью растения дикого типа,
(x) вегетативные части растений имеют уменьшенное соотношение углерод:азот по сравнению с соответствующей вегетативной частью растения дикого типа,
(xi) листья растения вида Sorghum и/или Zea mays имеют увеличенную способность к фотосинтезу по сравнению с соответствующим листом дикого типа,
(xii) вегетативные части растений имеют уменьшенное общее содержание пищевых волокон (TDF) по сравнению с соответствующей вегетативной частью растения дикого типа,
(xiii) вегетативные части растений имеют увеличенное содержание углерода по сравнению с соответствующей вегетативной частью растения дикого типа,
(xiv) вегетативные части растений имеют увеличенное содержание полипептида фактора транскрипции по сравнению с соответствующей вегетативной частью растения дикого типа, где полипептид фактора транскрипции выбран из группы, состоящей из Wrinkled 1 (WRI1), листовой семядоли 1 (LEC1), LEC1-подобного, листовой семядоли 2 (LEC2), BABY BOOM (BBM), FUS3, ABI3, ABI4, ABI5, Dof4 и Dof11, или из группы, состоящей из MYB73, bZIP53, AGL15, MYB115, MYB118, TANMEI, WUS, GFR2a1, GFR2a2 и PHR1,
(xv) вегетативные части растений имеют увеличенное содержание полипептида ацилтрансферазы жирных кислот по сравнению с соответствующей вегетативной частью растения дикого типа, где ацилтрансфераза представляет собой диацилглицерин-ацилтрансферазу (DGAT) и/или фосфолипид:диацилглицерин-ацилтрансферазу (PDAT),
(xvi) вегетативные части растений имеют уменьшенное содержание полипептида TAG-липазы по сравнению с соответствующей вегетативной частью растения дикого типа,
(xvii) вегетативные части растений имеют уменьшенное содержание полипептида тригалактозилдиацилглицерина (TGD) по сравнению с соответствующей вегетативной частью растения дикого типа,
(xviii) вегетативные части растений имеют увеличенное содержание полипептида покрытия масляных телец (OBC) или ассоциированного с липидными капельками полипептида (LDAP) по сравнению с соответствующей вегетативной частью растения дикого типа,
(xix) вегетативные части растений имеют увеличенное общее содержание белка по сравнению с соответствующей вегетативной частью растения дикого типа,
(xx) вегетативные части растений имеют увеличенное содержание хлорофилла по сравнению с соответствующей вегетативной частью растения дикого типа,
(xxi) вегетативные части растений имеют увеличенное содержание энергии в пересчете на массу по сравнению с соответствующей вегетативной частью растения дикого типа,
(xxii) вегетативные части растений имеют увеличенное содержание фосфолипидов и/или галактолипидов, предпочтительно, увеличенное содержание моногалактозил-диглицерида (MDGD) и/или увеличенное содержание дигалактозил-диглицерида (DGDG), по сравнению с соответствующей вегетативной частью растения дикого типа,
(xxiii) соотношение жирных кислот, этерифицированных в форме TAG, к жирным кислотам в форме липидов, отличных от TAG, составляет приблизительно 4, приблизительно 3,5, приблизительно 3, или приблизительно 2,5,
(xxiv) по меньшей мере один другой кормовой ингредиент содержит одно или несколько, или все из: съедобных питательных макровеществ, витаминов, минералов (таких как кальций, фосфор, магний и сера), сена, такого как люцерновое сено, пивной дробины, жмыховой муки (из канолы или сои), хлопчатника, мелассы, дополнительных аминокислот (таких как лизин и метионин) небелковых источников азота (таких как мочевина),
(xxv) период времени составляет между одной неделей и 52 неделями,
(xxvi) органические кислоты включают уксусную кислоту, пропионовую кислоту или масляную кислоту, или любую их комбинацию,
(xxvii) пищевой продукт представляет собой силос, гранулы или сено, и
(xxviii) вегетативные части растений хранят в течение некоторого периода времени перед смешиванием по меньшей мере с одним другим кормовым ингредиентом,
(xxiv) TTQ составляет приблизительно 0,1 или приблизительно 0,2 или приблизительно 0,3, или приблизительно 0,4 или приблизительно 0,5, или приблизительно 0,6, или приблизительно 0,65, или приблизительно 0,7, или приблизительно 0,75, или приблизительно 0,8, или приблизительно 0,81, или приблизительно 0,82, или приблизительно 0,83, или приблизительно 0,84, или приблизительно 0,85, или приблизительно 8,6, или приблизительно 8,7, или приблизительно 8,8, или приблизительно 8,9, или приблизительно 0,9, или приблизительно 0,91, или приблизительно 0,92, или приблизительно 0,93, или приблизительно 0,94, или приблизительно 0,95,
в каждом случае, когда соответствующую часть растения дикого типа собирают из растения вида Sorghum и/или Zea mays дикого типа на такой же стадии роста.
В следующем варианте осуществления вышеуказанных аспектов, используют один или несколько, или все из следующих признаков:
(i) вегетативные части растений представляют собой листья и/или стебли, или их части, имеющие одно или несколько из увеличенного содержания углерода, увеличенного содержания энергии, увеличенного содержания растворимого белка и увеличенного содержания азота, каждое в пересчете на массу по сравнению с соответствующим листом или стеблем дикого типа, или их частями, из растения вида Sorghum и/или Zea mays дикого типа на такой же стадии роста,
(ii) общее содержание жирных кислот в вегетативных частях растений составляет по меньшей мере приблизительно 8%, по меньшей мере приблизительно 9%, по меньшей мере приблизительно на 10%, по меньшей мере приблизительно 11%, по меньшей мере приблизительно 12%, по меньшей мере приблизительно 15%, по меньшей мере приблизительно 20%, по меньшей мере приблизительно 25%, по меньшей мере приблизительно 30%, по меньшей мере приблизительно 35%, по меньшей мере приблизительно 40%, по меньшей мере приблизительно 45%, по меньшей мере приблизительно 50%, по меньшей мере приблизительно 55%, по меньшей мере приблизительно 60%, по меньшей мере приблизительно 65%, по меньшей мере приблизительно 70%, между приблизительно 6% и приблизительно 20%, между 8% и 75%, между 10% и 75%, между 11% и 75%, между приблизительно 15% и 75%, между приблизительно 20% и 75%, между приблизительно 30% и 75%, между приблизительно 40% и 75%, между приблизительно 50% и 75%, между приблизительно 60% и 75%, или между приблизительно 25% и 50% (масс./масс. сухой массы),
(iii) жирные кислоты, этерифицированные в форме TAG, в вегетативных частях растений составляют по меньшей мере приблизительно 1%, по меньшей мере приблизительно 2%, по меньшей мере приблизительно 3%, по меньшей мере приблизительно 4%, по меньшей мере приблизительно 5%, по меньшей мере приблизительно 6%, по меньшей мере приблизительно 7%, по меньшей мере приблизительно 8%, по меньшей мере приблизительно 9%, по меньшей мере приблизительно 10%, по меньшей мере приблизительно 11%, по меньшей мере приблизительно 12%, по меньшей мере приблизительно 15%, по меньшей мере приблизительно 20%, по меньшей мере приблизительно 25%, по меньшей мере приблизительно 30%, по меньшей мере приблизительно 35%, по меньшей мере приблизительно 40%, по меньшей мере приблизительно 45%, по меньшей мере приблизительно 50%, по меньшей мере приблизительно 55%, по меньшей мере приблизительно 60%, по меньшей мере приблизительно 65%, по меньшей мере приблизительно 70%, между приблизительно 6% и приблизительно 20%, между 8% и 75%, между 10% и 75%, между 11% и 75%, между приблизительно 15% и 75%, между приблизительно 20% и 75%, между приблизительно 30% и 75%, между приблизительно 40% и 75%, между приблизительно 50% и 75%, между приблизительно 60% и 75%, или между приблизительно 25% и 50% (масс./масс. сухой массы),
(iv) вегетативные части растений имеют увеличенное содержание полипептида WRI1, увеличенное содержание полипептида DGAT и уменьшенное содержание полипептида SDP1, каждое по сравнению с соответствующей вегетативной частью растения дикого типа,
(v) вегетативные части растений имеют увеличенное содержание полипептида WRI1, увеличенное содержание полипептида DGAT, и увеличенное содержание полипептида LEC2, каждое по сравнению с соответствующей вегетативной частью растения дикого типа,
(vi) вегетативные части растений имеют увеличенное содержание полипептида PDAT или DGAT, уменьшенное содержание полипептида TGD и уменьшенное содержание полипептида SDP1, каждое по сравнению с соответствующей вегетативной частью растения дикого типа, и
(vii) вегетативные части растений имеют уменьшенное содержание полипептида TAG-липазы, такого как полипептид SDP1, уменьшенное содержание полипептида TGD, такого как полипептид TGD5, и необязательно, уменьшенное содержание полипептида TST, такого как полипептид TST1, каждое по сравнению с соответствующей вегетативной частью растения дикого типа.
В одном варианте осуществления, вегетативные части растений имеют увеличенное содержание одного или нескольких полипептидов метаболизма сахарозы, выбранных из группы, состоящей из инвертазы и полипептида для транспорта сахарозы. Инвертаза может представлять собой вакуолярную инвертазу или цитозольную инвертазу, и полипептид для транспорта сахарозы может представлять собой, например, SUS4 или SUT2, естественным образом локализованный на вакуолярной мембране.
В другом аспекте настоящее изобретение относится к способу кормления животного, включающему предоставление животному вегетативных частей растений из растения вида Sorghum и/или Zea mays, имеющих общее содержание жирных кислот (TFA), включающее жирные кислоты, этерифицированные в форме триацилглицеринов (TAG), и жирные кислоты в форме липидов, отличных от TAG, где вегетативные части растений имеют содержание TFA приблизительно 5% (масс./масс. сухой массы), предпочтительно, между приблизительно 6% и приблизительно 20%.
В одном варианте осуществления вышеуказанного аспекта, вегетативные части растений имеют TTQ между 0,01 и 0,6. В одном варианте осуществления, вегетативные части растений имеют TTQ между 0,01 и 0,55 или между 0,01 и 0,5, или приблизительно 0,1, или приблизительно 0,2 или приблизительно 0,3, или приблизительно 0,4, или приблизительно 0,5. Предпочтительно, TTQ составляет между 0,60 и 0,84 или между 0,84 и 0,95.
В другом аспекте настоящее изобретение относится к способу кормления животного, включающему предоставление животному вегетативных частей растений из растения вида Sorghum и/или Zea mays, имеющих общее содержание жирных кислот, включающее жирные кислоты, этерифицированные в форме триацилглицеринов (TAG), и жирные кислоты в форме липидов, отличных от TAG, где вегетативные части растений имеют общее содержание TAG приблизительно 6% (масс./масс. сухой массы), предпочтительно, между приблизительно 6% и приблизительно 20%, и предпочтительно, имеют соотношение жирных кислот, этерифицированных в форме TAG, к жирным кислотам в форме липидов, отличных от TAG, составляющее между 20:1 и 1,5:1 или между 5:1 и 2:1.
В одном варианте осуществления двух вышеуказанных аспектов, используют один или несколько, или все из следующих признаков:
(i) вегетативные части растений содержатся в растении вида Sorghum и/или Zea mays, растущем в поле,
(ii) вегетативные части растений собирают из растения вида Sorghum и/или Zea mays, и/или смешивают по меньшей мере с одним другим кормовым ингредиентом,
(iii) вегетативные части растений переработаны после сбора, предпочтительно, посредством рубки, нарезки, сушки, прессования или гранулирования частей растений, в форму, более пригодную для употребления животным,
(iv) собранные части растений хранили в условиях уменьшенного содержания кислорода в течение такого периода времени, что по меньшей мере некоторые из углеводов в частях растений подверглись ферментации до органических кислот, до предоставления животному, и
(v) собранные части растений хранят в течение некоторого периода времени между их сбором и предоставлением животному.
В следующем варианте осуществления двух вышеуказанных аспектов, животное употребляет внутрь увеличенное количество азота, белка, углерода и/или энергетического потенциала относительно того, когда животное употребляет внутрь такое же количество, в пересчете на сухую массу, соответствующего пищевого продукта, полученного с использованием эквивалентного количества растения дикого типа вида Sorghum и/или Zea mays, или его частей.
В следующем варианте осуществления двух вышеуказанных аспектов, способ дополнительно отличается одним или несколькими признаками, как описано в контексте первого или второго аспектов изобретения.
Два вышеуказанных аспекта описаны применительно к растению вида Sorghum и/или Zea mays. Однако, они не предназначены для ограничения описанных способов использованием только растения вида Sorghum и/или Zea mays. Подразумевают, что любое пригодное растение можно использовать в этих способов.
В следующем аспекте, настоящее изобретение относится a пищевому продукту для животного, содержащему собранные вегетативные части растений из растения вида Sorghum и/или Zea mays, имеющие общее содержание жирных кислот (TFA), включающее жирные кислоты, этерифицированные в форме триацилглицеринов (TAG), и жирные кислоты в форме липидов, отличных от TAG, где вегетативные части растений имеют содержание TFA приблизительно 5% (масс./масс. сухой массы), предпочтительно, между приблизительно 6% и приблизительно 20%, где
(i) собранные части растений смешаны по меньшей мере с одним другим кормовым ингредиентом,
(ii) собранные части растений упакованы в тюки после сбора,
(iii) собранные части растений переработаны, предпочтительно, посредством рубки, нарезки, сушки, прессования или гранулирования частей растений, в форму, пригодную для употребления животным, и
(iv) собранные части растений хранили в условиях уменьшенного содержания кислорода в течение такого периода времени, что по меньшей мере некоторые из углеводов в частях растений подверглись ферментации до органических кислот.
В одном варианте осуществления, вегетативные части растений имеют TTQ между 0,01 и 0,6. В одном варианте осуществления, вегетативные части растений имеют TTQ между 0,01 и 0,55 или между 0,01 и 0,5, или приблизительно 0,1, или приблизительно 0,2, или приблизительно 0,3, или приблизительно 0,4, или приблизительно 0,5. Предпочтительно, TTQ составляет между 0,60 и 0,84 или между 0,84 и 0,95.
В другом аспекте настоящее изобретение относится a пищевому продукту для животного, содержащему собранные вегетативные части растений из растения вида Sorghum и/или Zea mays, имеющие общее содержание жирных кислот, включающее жирные кислоты, этерифицированные в форме триацилглицеринов (TAG), и жирные кислоты в форме липидов, отличных от TAG, где вегетативные части растений имеют общее содержание TAG приблизительно 6% (масс./масс. сухой массы), предпочтительно, между приблизительно 6% и приблизительно 20%, и предпочтительно, имеют соотношение жирных кислот, этерифицированных в форме TAG, к жирным кислотам в форме липидов, отличных от TAG, составляющее между 20:1 и 1,5:1 или между 5:1 и 2:1, где
(i) собранные части растений смешаны по меньшей мере с одним другим кормовым ингредиентом,
(ii) собранные части растений упакованы в тюки после сбора,
(iii) собранные части растений переработаны, предпочтительно, посредством рубки, нарезки, сушки, прессования или гранулирования частей растений, в форму, пригодную для употребления животным, и
(iv) собранные части растений хранили в условиях уменьшенного содержания кислорода в течение такого периода времени, что по меньшей мере некоторые из углеводов в частях растений подверглись ферментации до органических кислот.
В одном варианте осуществления двух вышеуказанных аспектов, пищевой продукт представляет собой силос, гранулы или сено.
В следующем варианте осуществления двух вышеуказанных аспектов, пищевой продукт дополнительно отличается одним или несколькими признаками, как описано в контексте первого или второго аспектов изобретения.
Два вышеуказанных аспекта описаны применительно к растению вида Sorghum и/или Zea mays. Однако, они не предназначены для ограничения описанных пищевых продуктов содержанием вегетативных частей растений из растения вида Sorghum и/или Zea mays. Подразумевают, что пищевые продукты относятся, в более общем смысле, к пищевым продуктам, содержащим вегетативные части растений из любого пригодного растения.
В другом аспекте настоящее изобретение относится к клетке, предпочтительно, к клетке вида Sorghum или Zea mays, отличной от клетки семян, имеющей общее содержание жирных кислот (TFA), включающее жирные кислоты, этерифицированные в форме триацилглицеринов (TAG), и жирные кислоты в форме липидов, отличных от TAG, где клетка имеет содержание TFA приблизительно 5% (масс./масс. сухой массы), предпочтительно, между приблизительно 6% и приблизительно 20%.
В одном варианте осуществления, общее содержание жирных кислот в клетке имеет TTQ между 0,01 и 0,6. В одном варианте осуществления, общее содержание жирных кислот в клетке имеет TTQ между 0,01 и 0,55 или между 0,01 и 0,5, или приблизительно 0,1, или приблизительно 0,2, или приблизительно 0,3, или приблизительно 0,4, или приблизительно 0,5. Предпочтительно, TTQ составляет между 0,60 и 0,84 или между 0,84 и 0,95.
В одном варианте осуществления, содержание TFA в клетке включает содержание олеиновой кислоты, которое увеличено по меньшей мере на 2% или по меньшей мере на 3% относительно содержания олеиновой кислоты в соответствующей клетке дикого типа.
В одном варианте осуществления, содержание TFA в клетке включает содержание пальмитиновой кислоты, которое увеличено по меньшей мере на 2% или по меньшей мере на 3% относительно содержания пальмитиновой кислоты в соответствующей клетке дикого типа.
В одном варианте осуществления, содержание TFA в клетке включает содержание α-линолеиновой кислоты (ALA), которое уменьшено по меньшей мере на 2% или по меньшей мере на 3% относительно содержания ALA в соответствующей клетке дикого типа.
В одном варианте осуществления, клетка находится в вегетативной части растения и имеет содержание TAG по меньшей мере приблизительно 1%, по меньшей мере приблизительно 2%, по меньшей мере приблизительно 3%, по меньшей мере приблизительно 4%, по меньшей мере приблизительно 5%, по меньшей мере приблизительно 6%, по меньшей мере приблизительно 7%, по меньшей мере приблизительно 8%, по меньшей мере приблизительно на 10%, по меньшей мере приблизительно 11%, по меньшей мере приблизительно 12%, по меньшей мере приблизительно 15%, по меньшей мере приблизительно 20%, по меньшей мере приблизительно 25%, по меньшей мере приблизительно 30%, по меньшей мере приблизительно 35%, по меньшей мере приблизительно 40%, по меньшей мере приблизительно 45%, по меньшей мере приблизительно на 50%, по меньшей мере приблизительно 55%, по меньшей мере приблизительно 60%, по меньшей мере приблизительно 65%, по меньшей мере приблизительно 70%, между приблизительно 6% и приблизительно 20%, между 8% и 75%, между 10% и 75%, между 11% и 75%, между приблизительно 15% и 75%, между приблизительно 20% и 75%, между приблизительно 30% и 75%, между приблизительно 40% и 75%, между приблизительно 50% и 75%, между приблизительно 60% и 75%, или между приблизительно 25% и 50% (масс./масс. сухой массы).
В следующем варианте осуществления, клетка получена из листа или стебля растения, или находится в листе или стебле растения, до цветения растения, и клетка имеет содержание TFA и/или общее содержание неполярных жирных кислот по меньшей мере приблизительно 6%, по меньшей мере приблизительно 7%, по меньшей мере приблизительно 8%, по меньшей мере приблизительно 10%, по меньшей мере приблизительно 11%, между 8% и 15%, или между 9% и 12% масс., предпочтительно, между приблизительно 6% и приблизительно 20%.
В другом аспекте настоящее изобретение относится к клетке, предпочтительно, к клетке вида Sorghum или Zea mays, отличной от клетки семян, имеющей общее содержание жирных кислот, включающее жирные кислоты, этерифицированные в форме триацилглицеринов (TAG), и жирные кислоты в форме липидов, отличных от TAG, где клетка имеет общее содержание TAG приблизительно 6% (масс./масс. сухой массы), предпочтительно, между приблизительно 6% и приблизительно 20%, и имеет соотношение жирных кислот, этерифицированных в форме TAG, к жирным кислотам в форме липидов, отличных от TAG, составляющее между 20:1 и 1,5:1 или между 5:1 и 2:1.
В другом аспекте настоящее изобретение относится к клетке, предпочтительно, к клетке вида Sorghum или Zea mays, имеющей увеличенное содержание полипептида WRI1, увеличенное содержание полипептида DGAT и уменьшенное содержание полипептида SDP1, каждое по сравнению с соответствующей клеткой дикого типа.
В другом аспекте настоящее изобретение относится к клетке, предпочтительно, к клетке вида Sorghum или Zea mays, имеющей увеличенное содержание полипептида WRI1, увеличенное содержание полипептида DGAT, и увеличенное содержание полипептида LEC2, каждое по сравнению с соответствующей клеткой дикого типа.
В другом аспекте настоящее изобретение относится к клетке, предпочтительно, к клетке вида Sorghum или Zea mays, имеющей увеличенное содержание полипептида PDAT или DGAT, уменьшенное содержание полипептида TGD, предпочтительно, полипептида TGD5, и уменьшенное содержание полипептида SDP1, каждое по сравнению с соответствующей клеткой дикого типа.
В другом аспекте настоящее изобретение относится к клетке, предпочтительно, к клетке вида Sorghum или Zea mays, имеющей уменьшенное содержание TAG-липазы, такой как TAG-липаза SDP1, уменьшенное содержание полипептида TGD, такого как полипептид TGD5, и необязательно уменьшенное содержание полипептида TST, такого как полипептид TST1, где каждое уменьшение присутствует по сравнению с соответствующей клеткой дикого типа.
В одном варианте осуществления вышеуказанных аспектов, относящихся к клетке по изобретению, используют один или несколько, или все из следующих признаков:
(i) клетка находится в вегетативной части растения, собранной из растения вида Sorghum и/или Zea mays, между временем первого цветения растения и первого созревания семян,
(ii) клетка представляет собой клетку растения Sorghum bicolor,
(iii) клетка находится в листе или стебле, или его части,
(iv) клетка имеет общее содержание липидов приблизительно 8% или приблизительно 10% масс.,
(v) общее содержание жирных кислот в клетке включает содержание олеиновой кислоты, которое увеличено по меньшей мере на 2% или по меньшей мере на 3% относительно содержания олеиновой кислоты в соответствующей клетке дикого типа,
(vi) общее содержание жирных кислот в клетке включает содержание пальмитиновой кислоты, которое увеличено по меньшей мере на 2% или по меньшей мере на 3% относительно содержания пальмитиновой кислоты в соответствующей клетке дикого типа,
(vii) общее содержание жирных кислот в клетке включает содержание α-линолеиновой кислоты (ALA), которое уменьшено по меньшей мере на 2% или по меньшей мере на 3% относительно содержания ALA в соответствующей клетке дикого типа,
(viii) клетка имеет увеличенное содержание растворимого белка по сравнению с соответствующей клеткой дикого типа,
(ix) клетка имеет увеличенное содержание азота по сравнению с соответствующей клеткой дикого типа,
(x) клетка имеет уменьшенное соотношение углерод:азот по сравнению с соответствующей клеткой дикого типа,
(xi) клетка имеет увеличенную способность к фотосинтезу по сравнению с соответствующей клеткой дикого типа,
(xii) клетка имеет уменьшенное содержание крахмала и/или общее содержание пищевых волокон (TDF) по сравнению с соответствующей клеткой дикого типа,
(xiii) клетка имеет увеличенное содержание углерода по сравнению с соответствующей клеткой дикого типа,
(xiv) клетка имеет увеличенное содержание полипептида фактора транскрипции по сравнению с соответствующей клеткой дикого типа, где полипептид фактора транскрипции выбран из группы, состоящей из Wrinkled 1 (WRI1), листовой семядоли 1 (LEC1), LEC1-подобного, листовой семядоли 2 (LEC2), BABY BOOM (BBM), FUS3, ABI3, ABI4, ABI5, Dof4 и Dof11, или из группы, состоящей из MYB73, bZIP53, AGL15, MYB115, MYB118, TANMEI, WUS, GFR2a1, GFR2a2 и PHR1,
(xv) клетка имеет увеличенное содержание полипептида ацилтрансферазы жирных кислот по сравнению с соответствующей клеткой дикого типа, где ацилтрансфераза представляет собой диацилглицерин-ацилтрансферазу (DGAT) и/или фосфолипид:диацилглицерин-ацилтрансферазу (PDAT),
(xvi) клетка имеет уменьшенное содержание полипептида TAG-липазы по сравнению с соответствующей клеткой дикого типа,
(xvii) клетка имеет уменьшенное содержание полипептида тригалактозилдиацилглицерина (TGD) по сравнению с соответствующей клеткой дикого типа,
(xviii) клетка имеет увеличенное содержание полипептида покрытия масляных телец (OBC) или ассоциированного с липидными капельками полипептида (LDAP) по сравнению с соответствующей клеткой дикого типа,
(xix) клетка имеет увеличенное общее содержание белка по сравнению с соответствующей клеткой дикого типа,
(xx) клетка имеет увеличенное содержание хлорофилла по сравнению с соответствующей клеткой дикого типа,
(xxi) клетка имеет увеличенное содержание энергии в пересчете на массу по сравнению с соответствующей клеткой дикого типа,
(xxii) клетка имеет увеличенное содержание фосфолипидов и/или галактолипидов по сравнению с соответствующей клеткой дикого типа, предпочтительно, увеличенное содержание MDGD и/или увеличенное содержание DGDG,
(xxiii) TTQ составляет приблизительно 0,1 или приблизительно 0,2, или приблизительно 0,3, или приблизительно 0,4 или приблизительно 0,5, или приблизительно 0,6, или приблизительно 0,65, или приблизительно 0,7, или приблизительно 0,75, или приблизительно 0,8, или приблизительно 0,81, или приблизительно 0,82, или приблизительно 0,83, или приблизительно 0,84, или приблизительно 0,85, или приблизительно 8,6, или приблизительно 8,7, или приблизительно 8,8, или приблизительно 8,9, или приблизительно 0,9, или приблизительно 0,91, или приблизительно 0,92, или приблизительно 0,93, или приблизительно 0,94, или приблизительно 0,95, и
(xxiv) соотношение жирных кислот, этерифицированных в форме TAG, к жирным кислотам в форме липидов, отличных от TAG, составляет между 20:1 и 1,5:1 или между 5:1 и 2:1, или приблизительно 4, приблизительно 3,5, приблизительно 3, или приблизительно 2,5.
В одном варианте осуществления вышеуказанных аспектов, вегетативные части растений или клетка по изобретению содержат одно или оба из
a) первого экзогенного полинуклеотида, кодирующего полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в вегетативных частях растений или клетке, предпочтительно, полипептид WRI1, и
b) второго экзогенного полинуклеотида, кодирующего полипептид, вовлеченный в биосинтез одного или нескольких неполярных липидов, предпочтительно, DGAT и/или PDAT, и в каждом случае, любое одно или два, или три, или все четыре из
c) первой генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в катаболизм триацилглицеринов (TAG) в вегетативных частях растений или клетке, предпочтительно, TAG-липазы SDP1, по сравнению с соответствующими вегетативной частью растения или клеткой, лишенными генетической модификации,
d) третьего экзогенного полинуклеотида, кодирующего полипептид, который увеличивает экспорт жирных кислот из пластид в вегетативных частях растений или клетке, по сравнению с соответствующими вегетативной частью растения или клеткой, лишенными третьего экзогенного полинуклеотида, предпочтительно, полипептид ацил-ACP-тиоэстеразы,
e) четвертого экзогенного полинуклеотида, кодирующего второй полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в вегетативных частях растений или клетке, предпочтительно, полипептид LEC2, и
f) второй генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в импорт жирных кислот в пластиды в вегетативных частях растений или клетке по сравнению с соответствующими вегетативной частью растения или клеткой, лишенными второй генетической модификации, предпочтительно, полипептид TGD,
где каждый экзогенный полинуклеотид является функционально связанным с промотором, способным управлять экспрессией полинуклеотида в вегетативных частях растений или клетке.
В одном варианте осуществления вышеуказанных аспектов, вегетативные части растений или клетка дополнительно содержат одно или оба из
a) пятого экзогенного полинуклеотида, кодирующего полипептид покрытия масляных телец (OBC) или ассоциированный с липидными капельками белок (LDAP), и
b) третьей генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в продукцию диацилглицерина (DAG) в пластиде, по сравнению с соответствующими вегетативной частью растения или клеткой, лишенными третьей генетической модификации.
В альтернативном варианте осуществления вышеуказанных аспектов, вегетативные части растений или клетка содержат
a) первый экзогенный полинуклеотид, кодирующий полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в вегетативных частях растений или клетке, предпочтительно, полипептид WRI1,
b) второй экзогенный полинуклеотид, кодирующий полипептид, вовлеченный в биосинтез одного или нескольких неполярных липидов, предпочтительно, DGAT и/или PDAT, и любое одно или два, или все три из
c) первой генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в катаболизм триацилглицеринов (TAG) в вегетативных частях растений или клетке, по сравнению с соответствующими вегетативной частью растения или клеткой, лишенными генетической модификации, предпочтительно, TAG-липазы SDP1,
d) третьего экзогенного полинуклеотида, кодирующего полипептид, который увеличивает экспорт жирных кислот из пластид в вегетативных частях растений или клетке по сравнению с соответствующими вегетативной частью растения или клеткой, лишенными третьего экзогенного полинуклеотида, предпочтительно, полипептид ацил-ACP-тиоэстеразы, и
e) четвертого экзогенного полинуклеотида, кодирующего второй полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в вегетативных частях растений или клетке, предпочтительно, полипептид LEC2,
где каждый экзогенный полинуклеотид является функционально связанным с промотором, способным управлять экспрессией полинуклеотида в вегетативных частях растений или клетке.
В одном варианте осуществления вышеуказанного аспекта, вегетативные части растений или клетка дополнительно содержат одно или несколько, или все из
a) пятого экзогенного полинуклеотида, кодирующего полипептид покрытия масляных телец (OBC) или ассоциированный с липидными капельками белок (LDAP),
b) второй генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в импорт жирных кислот в пластиды в вегетативных частях растений или клетке, по сравнению с соответствующими вегетативной частью растения или клеткой, лишенными второй генетической модификации, и
c) третьей генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в продукцию диацилглицерина (DAG) в пластиде, по сравнению с соответствующими вегетативной частью растения или клеткой, лишенными третьей генетической модификации.
В следующем варианте осуществления вышеуказанных аспектов, вегетативные части растений или клетка содержат первый экзогенный полинуклеотид, кодирующий полипептид WRI1, второй экзогенный полинуклеотид, кодирующий полипептид DGAT, и уменьшенное содержание полипептида TAG-липазы и/или уменьшенное содержание полипептида TGD, по сравнению с соответствующей вегетативной частью растения или клеткой дикого типа, где каждый экзогенный полинуклеотид является функционально связанным с промотором, способным управлять экспрессией полинуклеотида в вегетативных частях растений или клетке.
В другом варианте осуществления вышеуказанных аспектов, вегетативные части растений или клетка содержат экзогенный полинуклеотид, кодирующий полипептид PDAT или DGAT, увеличенное содержание полипептида PDAT или DGAT, уменьшенное содержание полипептида TGD и уменьшенное содержание полипептида TAG-липазы, каждое по сравнению с соответствующей вегетативной частью растения или клеткой дикого типа, где экзогенный полинуклеотид является функционально связанным с промотором, способным управлять экспрессией полинуклеотида в вегетативных частях растений или клетке.
В другом варианте осуществления вышеуказанных аспектов, вегетативные части растений или клетка содержат уменьшенное содержание TAG-липазы, такой как TAG-липаза SDP1, уменьшенное содержание полипептида TGD, такого как полипептид TGD5, и необязательно, уменьшенное содержание полипептида TST, такого как полипептид TST1, где каждое уменьшение присутствует по сравнению с соответствующей вегетативной частью растения или клеткой дикого типа.
В следующем варианте осуществления вышеуказанных аспектов, клетка происходит из или находится в вегетативной части растения вида Sorghum и/или Zea mays.
В следующем варианте осуществления вышеуказанных аспектов, используют один или несколько, или все из следующих признаков:
i) вегетативные части растений или клетка имеют увеличенный синтез общих жирных кислот по сравнению с соответствующей вегетативной частью растения или клеткой, лишенной первого экзогенного полинуклеотида, или уменьшенный катаболизм общих жирных кислот по сравнению с соответствующей вегетативной частью растения или клеткой, лишенной первого экзогенного полинуклеотида, или оба, таким образом, что они имеют увеличенный уровень общих жирных кислот по сравнению с соответствующей вегетативной частью растения или клеткой, лишенной первого экзогенного полинуклеотида,
ii) вегетативные части растений или клетка имеют увеличенную экспрессию и/или активность ацилтрансферазы жирных ацилов, катализирующей синтез TAG, DAG или MAG, предпочтительно, TAG, по сравнению с соответствующей вегетативной частью растения или клеткой, имеющей первый экзогенный полинуклеотид и лишенной экзогенного полинуклеотида, который кодирует полипептид, вовлеченный в биосинтез одного или нескольких неполярных липидов,
iii) вегетативные части растений или клетка имеют уменьшенную продукцию лизофосфатидиновой кислоты (LPA) из ацил-ACP и G3P в пластидах, по сравнению с соответствующей вегетативной частью растения или клеткой, имеющей первый экзогенный полинуклеотид и лишенной генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в продукцию диацилглицерина (DAG) в пластиде, в вегетативных частях растений или клетке,
iv) вегетативные части растений или клетка имеют измененное соотношение C16:3 и C18:3 жирных кислот в общем содержании жирных кислот и/или измененное содержание галактолипидов по сравнению с соответствующей вегетативной частью растения или клеткой, лишенной экзогенного полинуклеотида(полинуклеотидов) и/или генетической модификации(модификаций), предпочтительно, уменьшенное соотношение,
v) клетка находится в вегетативной части растения и имеет общее содержание неполярных липидов по меньшей мере приблизительно 8%, по меньшей мере приблизительно на 10%, по меньшей мере приблизительно 11%, по меньшей мере приблизительно 12%, по меньшей мере приблизительно 15%, по меньшей мере приблизительно 20%, по меньшей мере приблизительно 25%, по меньшей мере приблизительно 30%, по меньшей мере приблизительно 35%, по меньшей мере приблизительно 40%, по меньшей мере приблизительно 45%, по меньшей мере приблизительно 50%, по меньшей мере приблизительно 55%, по меньшей мере приблизительно 60%, по меньшей мере приблизительно 65%, по меньшей мере приблизительно 70%, между приблизительно 6% и приблизительно 20%, между 8% и 75%, между 10% и 75%, между 11% и 75%, между приблизительно 15% и 75%, между приблизительно 20% и 75%, между приблизительно 30% и 75%, между приблизительно 40% и 75%, между приблизительно 50% и 75%, между приблизительно 60% и 75%, или между приблизительно 25% и 50% (масс./масс. сухой массы),
vi) клетка находится в вегетативной части растения и имеет содержание TAG по меньшей мере приблизительно 1%, по меньшей мере приблизительно 2%, по меньшей мере приблизительно 3%, по меньшей мере приблизительно 4%, по меньшей мере приблизительно 5%, по меньшей мере приблизительно 6%, по меньшей мере приблизительно 7%, по меньшей мере приблизительно 8%, по меньшей мере приблизительно 10%, по меньшей мере приблизительно 11%, по меньшей мере приблизительно 12%, по меньшей мере приблизительно 15%, по меньшей мере приблизительно 20%, по меньшей мере приблизительно 25%, по меньшей мере приблизительно 30%, по меньшей мере приблизительно 35%, по меньшей мере приблизительно 40%, по меньшей мере приблизительно 45%, по меньшей мере приблизительно 50%, по меньшей мере приблизительно 55%, по меньшей мере приблизительно 60%, по меньшей мере приблизительно 65%, по меньшей мере приблизительно 70%, между приблизительно 6% и приблизительно 20%, между 8% и 75%, между 10% и 75%, между 11% и 75%, между приблизительно 15% и 75%, между приблизительно 20% и 75%, между приблизительно 30% и 75%, между приблизительно 40% и 75%, между приблизительно 50% и 75%, между приблизительно 60% и 75%, или между приблизительно 25% и 50% (масс./масс. сухой массы),
vii) полипептид фактора транскрипции выбран из группы, состоящей из Wrinkled 1 (WRI1), листовой семядоли 1 (LEC1), LEC1-подобного, листовой семядоли 2 (LEC2), BABY BOOM (BBM), FUS3, ABI3, ABI4, ABI5, Dof4 и Dof11,
viii) олеиновая кислота составляет по меньшей мере 20% (моль %), по меньшей мере 22% (моль %), по меньшей мере 30% (моль %), по меньшей мере 40% (моль %), по меньшей мере 50% (моль %), или по меньшей мере 60% (моль %), предпочтительно, приблизительно 65% (моль %) или между 20% и приблизительно 65% от общего содержания жирных кислот в вегетативных частях растений или клетке,
ix) неполярный липид в вегетативных частях растений или клетке содержит одну или несколько полиненасыщенных жирных кислот, выбранных из эйкозадиеновой кислоты (EDA), арахидоновой кислоты (ARA), стеаридоновой кислоты (SDA), эйкозатриеновой кислоты (ETE), эйкозатетраеновой кислоты (ETA), эйкозапентаеновой кислоты (EPA), докозапентаеновой кислоты (DPA), докозагексаеновой кислоты (DHA) или комбинации двух или более из них,
x) один или несколько, или все из промоторов выбраны из конститутивного промотора, такого как промотор гена убиквитина или промотор гена актина, тканеспецифического промотора, такого как специфический для листьев и/или стеблей промотор, регулируемого развитием промотора, такого как специфический для старения промотор, например, промотор SAG12, индуцибельного промотора или регулируемого суточным ритмом промотора,
xi) вегетативные части растений или клетка имеют общее содержание жирных кислот, уровень олеиновой кислоты в котором увеличен по меньшей мере на 2% или по меньшей мере на 3%, по сравнению с соответствующей вегетативной частью растения или клеткой, лишенной экзогенного полинуклеотида(полинуклеотидов) и/или генетической модификации(модификаций), и/или уровень α-линоленовой кислоты (ALA) в котором уменьшен по меньшей мере на 2% или по меньшей мере на 3%, по сравнению с соответствующей вегетативной частью растения или клеткой, лишенной экзогенного полинуклеотида(полинуклеотидов) и/или генетической модификации(модификаций),
xii) неполярный липид в вегетативных частях растений или клетке включает модифицированный уровень общих стеролов, предпочтительно, свободных (неэтерифицированных) стеролов, стероильных сложных эфиров, стероилгликозидов, по сравнению с неполярным липидом в соответствующей вегетативной части растения или клетке, лишенной экзогенного полинуклеотида(полинуклеотидов) и/или генетической модификации(модификаций),
xiii) уровень одного или нескольких неполярных липида(липидов) и/или общее содержание неполярных липидов в вегетативных частях растений или клетке по меньшей мере на 2% больше в пересчете на массу, чем в соответствующих вегетативных частях растений или клетке, содержащих экзогенные полинуклеотиды, кодирующие WRI1 Arabidposis thaliana (SEQ ID NO:21) и DGAT1 Arabidopsis thaliana (SEQ ID NO:1).
В следующем варианте осуществления вышеуказанных аспектов, используют один или несколько, или все из следующих признаков, когда это уместно;
i) полипептид, вовлеченный в биосинтез одного или нескольких неполярных липидов, представляет собой ацилтрансферазу жирных ацилов, вовлеченную в биосинтез TAG, DAG или моноацилглицерина (MAG) в клетке, такую как DGAT, PDAT, LPAAT, GPAT или MGAT, предпочтительно, DGAT или PDAT, или представляет собой полипептид PDCT или CPT, или полипептид PLC или PLD,
ii) полипептид, вовлеченный в катаболизм триацилглицеринов (TAG) в вегетативных частях растений или клетке, представляет собой липазу SDP1, полипептид Cgi58, ацил-CoA-оксидазу, такую как ACX1 или ACX2, или полипептид, вовлеченный в β-окисление жирных кислот в вегетативных частях растений или клетке, такой как пероксисомальный транспортер АТФ-связывающей кассеты PXA1, предпочтительно, липазу SDP1,
iii) полипептид покрытия масляных телец (OBC) представляет собой олеозин, такой как полиолеозин или калеозин, или ассоциированный с липидными капельками белок (LDAP),
iv) полипептид, который увеличивает экспорт жирных кислот из пластид в вегетативных частях растений или клетке, представляет собой тиоэстеразу C16 или C18 жирных кислот, такую как полипептид FATA или полипептид FATB, транспортер жирных кислот, такой как полипептид ABCA9, или длинноцепочечную ацил-CoA-синтетазу (LACS),
v) полипептид, вовлеченный в импорт жирных кислот в пластиды в вегетативных частях растений или клетке, представляет собой транспортер жирных кислот, или его субъединицу или регуляторный полипептид, предпочтительно, полипептид TGD, более предпочтительно, полипептид TGD5, и
vi) полипептид, вовлеченный в продукцию диацилглицерина (DAG) в пластиде представляет собой пластидную GPAT, пластидную LPAAT или пластидную PAP.
В одном варианте осуществления вышеуказанных аспектов, уровень или активность PDCT или CPT, или как PDCT, так и CPT, увеличены в вегетативной части растения, семени или клетке по изобретению, по сравнению с диким типом. В предпочтительном варианте осуществления, вегетативная часть растения, семя или клетка по изобретению содержит один или несколько экзогенных полинуклеотидов, кодирующих полипептид PDCT и/или CPT. Полипептид PDCT и/или CPT может являться эндогенным для вегетативной части растения, семени или клетки, т.е. происходящим из того же вида, но имеющим увеличенные уровень или активность, по сравнению с диким типом, или может являться гетерологичным для вида растения. Каждый экзогенный полинуклеотид является функционально связанным с промотором, экспрессируемым в вегетативной части растения, семени или клетке, для обеспечения увеличенного уровня или активности PDCT и/или CPT. В предпочтительном варианте осуществления, увеличенные уровень или активность PDCT и/или CPT обеспечивают увеличенную скорость перевода DAG в фосфатидилхолин (PC), или PC в DAG, или более предпочтительно, обоих из них. Вегетативная часть растения, семя или клетка, таким образом, имеет увеличенную продукцию DAG, полученного из PC, при этом DAG является доступным для получения TAG посредством активности DGAT. В более предпочтительном варианте осуществления, уровень TAG в вегетативной части растения, семени или клетке является увеличенным, по сравнению с соответствующей частью или клеткой, лишенной экзогенных полинуклеотидов.
Альтернативно, уровень или активность PDCT или CPT уменьшены, или уменьшены как для PDCT, так и для CPT, в вегетативной части растения, семени или клетки по изобретению, по сравнению с диким типом, например, посредством мутации в эндогенном гене, кодирующем фермент(ы), или посредством понижающей регуляции гена(генов), кодирующих фермент(ы), посредством молекулы РНК, уменьшающей их экспрессию. В этом варианте осуществления, присутствует уменьшенный перевод DAG, полученного посредством пути Кеннеди (de novo DAG), в PC, что приводит к увеличенному уровню de novo DAG, доступного для синтеза TAG в вегетативной части растения, семени или клетки.
В одном варианте осуществления вышеуказанных аспектов, уровень или активность фосфолипазы-C (PLC) или фосфолипазы-D (PLD), или как PLC, так и PLD, увеличены в вегетативной части растения, семени или клетке по изобретению, по сравнению с диким типом. В предпочтительном варианте осуществления, вегетативная часть растения, семя или клетка по изобретению содержит один или несколько экзогенных полинуклеотидов, кодирующих полипептид PLC и/или PLD. Полипептид PLC и/или PLD может являться эндогенным для вегетативной части растения или клетки, но имеющим увеличенные уровень или активность, по сравнению с диким типом, или может являться гетерологичным для вида растения. Экзогенный полинуклеотид является функционально связанным с промотором, экспрессируемым в вегетативной части растения или клетке, для обеспечения увеличенного уровня или активности PLC и/или PLD. В предпочтительном варианте осуществления, увеличенные уровень или активность PLC и/или PLD обеспечивают увеличенную скорость перевода PC в DAG и фосфохолин в случае PLC, или фосфатидную кислоту (PA) и холин в случае PLD. PA затем переводят в DAG под действием PAP. Вегетативная часть растения или клетка, таким образом, имеет увеличенное количество DAG, доступного для получения TAG посредством активности DGAT. В более предпочтительном варианте осуществления, уровень или активность TAG в вегетативной части растения или клетке является увеличенным, по сравнению с соответствующей частью или клеткой, лишенной экзогенных полинуклеотидов, кодирующих PLC и/или PLD. В предпочтительном варианте осуществления, увеличенные уровень или активность PLC и/или PLD присутствуют в комбинации с увеличенным уровнем или активностью PDCT и/или CPT, как описано выше.
В одном варианте осуществления вышеуказанных аспектов, уровень или активность PDAT являются увеличенными в вегетативной части растения или клетке по изобретению, по сравнению с диким типом. В предпочтительном варианте осуществления, вегетативная часть растения или клетка содержит один или несколько экзогенных полинуклеотидов, кодирующих полипептид PDAT. Полипептид PDAT может являться эндогенным для вегетативной части растения или клетки, но имеющим увеличенные уровень или активность, по сравнению с диким типом, или может являться гетерологичным для вида растения. Экзогенный полинуклеотид является функционально связанным с промотором, экспрессируемым в вегетативной части растения или клетке, для обеспечения увеличенного уровня или активности PDAT. Является желательным, чтобы увеличенные уровень или активность PDAT обеспечивали увеличенное получение TAG из DAG и PC. В предпочтительном варианте осуществления, увеличенные уровень или активность PDAT присутствуют в комбинации с увеличенными уровнем или активностью PDCT и/или CPT, или с увеличенными уровнем или активностью PLC и/или PLD, как описано выше.
В одном варианте осуществления вышеуказанных аспектов, уровень или активность двух различных DGAT увеличены в вегетативной части растения или клетке по изобретению по сравнению с диким типом. В предпочтительном варианте осуществления, вегетативная часть растения или клетка по изобретению содержит два экзогенных полинуклеотида, каждый из которых кодирует полипептид DGAT, где полипептиды являются различными. Один из полипептидов DGAT может являться эндогенным для вегетативной части растения или клетки, но имеющим увеличенные уровень или активность, по сравнению с диким типом, в то время как другой является гетерологичным, или оба DGAT являются гетерологичными для вида растения. Каждый из экзогенных полинуклеотидов является функционально связанным с промотором, экспрессируемым в вегетативной части растения или клетке, для обеспечения увеличенной экспрессии обоих DGAT. Является желательным, чтобы увеличенные уровень или активность двух DGAT обеспечивали увеличенную продукцию TAG, полученного из de novo DAG и из происходящего из PC DAG. В предпочтительном варианте осуществления, один из двух DGAT (первый DGAT) является более активным по отношению к de novo DAG, чем к происходящему из PC DAG, в то время как другой DGAT (второй DGAT) является более активным по отношению к происходящему из PC DAG, чем к de novo DAG. Такие различия в активности DGAT могут возникать в результате различной компартментализации или локализации двух DGAT в эндоплазматическом ретикулуме (ER) клетки. В одном варианте осуществления, один из двух DGAT происходит из одноклеточного организма, например, бактериальный DGAT или DGAT одноклеточной водоросли, такой как DGAT Chlamydomonas или его вариант. В предпочтительном варианте осуществления, первый DGAT происходит из вида растения, естественным образом образующего масло, имеющего низкий уровень полиненасыщенных жирных кислот (PUFA), например, менее чем 20% PUFA, или представляет собой его гомолог DGAT, по меньшей мере на 95% идентичный по аминокислотной последовательности, и второй DGAT происходит из масличной культуры, образующей относительно более высокий уровень PUFA, например, по меньшей мере 40% PUFA, или представляет собой его гомолог DGAT, по меньшей мере на 95% идентичный по аминокислотной последовательности. Например, первый DGAT может происходить из оливы, кокоса, пальмы или мангустана, и второй DGAT может происходить из Brassica, сои, хлопчатника или семени льна. Как правило, первый DGAT является более активным по отношению к de novo DAG, и второй DGAT является более активным по отношению к происходящему из PC DAG. В предпочтительном варианте осуществления, увеличенная активность двух DGAT присутствует в комбинации с увеличенными уровнем или активностью PDCT и/или CPT, или с увеличенными уровнем или активностью PLC и/или PLD, или с увеличенными уровнем или активностью PDAT, как описано выше.
В вышеуказанных вариантах осуществления, степень увеличения уровня или активности предпочтительно, составляет по меньшей мере 10% или по меньшей мере 20%, до максимального увеличения на 100% или 200%. Степень уменьшения уровня или активности предпочтительно, составляет по меньшей мере 10% или 20%, до максимального уменьшения на 90% или 95%, или даже на 100%.
В вышеуказанных вариантах осуществления, увеличенные уровень или активность приводят к увеличенному TTQ в общем содержании липидов в вегетативной части растения или клетке по изобретению и/или к увеличенному уровню TAG по сравнению с соответствующей вегетативной частью растения или клеткой, лишенной соответствующих экзогенных полинуклеотидов. В следующем варианте осуществления вышеуказанных аспектов, клетка по изобретению получена из листа или стебля растения, или находится в листе или стебле растения, до цветения растения, и клетка имеет содержание TFA и/или общее содержание неполярных жирных кислот по меньшей мере приблизительно 6%, по меньшей мере приблизительно 8%, по меньшей мере приблизительно 10%, по меньшей мере приблизительно 11%, между 8% и 15%, или между 9% и 12% масс., предпочтительно, между приблизительно 8% и приблизительно 20%.
В следующем варианте осуществления вышеуказанных аспектов, каждая генетическая модификация независимо представляет собой мутацию эндогенного гена, которая частично или полностью инактивирует ген, такую как точечная мутация, инсерция или делеция, или генетическая модификация представляет собой экзогенный полинуклеотид, кодирующий молекулу РНК, уменьшающую экспрессию эндогенного гена, где экзогенный полинуклеотид является функционально связанным с промотором, способным управлять экспрессией полинуклеотида в вегетативных частях растений или клетке.
В следующем аспекте, настоящее изобретение относится к растению, предпочтительно, растению вида Sorghum и/или Zea mays, или его части, где растение содержит вегетативную часть растения, общее содержание жирных кислот (TFA) в которой включает жирные кислоты, этерифицированные в форме триацилглицеринов (TAG), и жирные кислоты в форме липидов, отличных от TAG, где вегетативная часть растения имеет содержание TFA приблизительно 5% (масс./масс. сухой массы), предпочтительно, между приблизительно 6% и приблизительно 20%. В одном варианте осуществления, часть растения представляет собой семя или семена, полученные из растения, или семя или семена, высеянные для получения такого растения.
В другом аспекте настоящее изобретение относится к растению, предпочтительно, к растению вида Sorghum и/или Zea mays, или его части, где растение содержит вегетативную часть растения, общее содержание жирных кислот в которой включает жирные кислоты, этерифицированные в форме триацилглицеринов (TAG), и жирные кислоты в форме липидов, отличных от TAG, где вегетативная часть растения имеет общее содержание TAG приблизительно 6% (масс./масс. сухой массы), предпочтительно, между приблизительно 6% и приблизительно 20%, и предпочтительно, имеет соотношение жирных кислот, этерифицированных в форме TAG, к жирным кислотам в форме липидов, отличных от TAG, составляющее между 20:1 и 1,5:1 или между 5:1 и 2:1.
В другом аспекте настоящее изобретение относится к растению, предпочтительно, растению вида Sorghum и/или Zea mays, или его части, где растение содержит вегетативную часть растения, имеющую увеличенное содержание полипептида WRI1, увеличенное содержание полипептида DGAT и уменьшенное содержание полипептида SDP1, каждое по сравнению с соответствующей вегетативной частью растения дикого типа.
В другом аспекте настоящее изобретение относится к растению, предпочтительно, растению вида Sorghum и/или Zea mays, или его части, где растение содержит вегетативную часть растения, имеющую увеличенное содержание полипептида WRI1, увеличенное содержание полипептида DGAT, и увеличенное содержание полипептида LEC2, каждое по сравнению с соответствующей вегетативной частью растения дикого типа.
В другом аспекте настоящее изобретение относится к растению, предпочтительно, растению вида Sorghum и/или Zea mays, или его части, где растение содержит вегетативную часть растения, имеющую увеличенное содержание полипептида PDAT или DGAT, уменьшенное содержание полипептида TGD, предпочтительно, полипептида TGD5, и уменьшенное содержание полипептида SDP1, каждое по сравнению с соответствующей вегетативной частью растения дикого типа.
В другом аспекте настоящее изобретение относится к растению, предпочтительно, растению вида Sorghum и/или Zea mays, или его части, содержащей вегетативную часть растения, имеющую уменьшенное содержание TAG-липазы, такой как TAG-липаза SDP1, уменьшенное содержание полипептида TGD, такого как полипептид TGD5, и необязательно, уменьшенное содержание полипептида TST, такого как полипептид TST1, где каждое уменьшение присутствует по сравнению с соответствующей вегетативной частью растения дикого типа.
В другом аспекте настоящее изобретение относится к растению, предпочтительно, растению вида Sorghum и/или Zea mays, или его части, где растение содержит вегетативную часть, имеющую увеличенный уровень или активность PDCT и/или CPT по сравнению с диким типом. В одном варианте осуществления, растение или его часть содержат один или несколько экзогенных полинуклеотидов, кодирующих полипептид PDCT и/или CPT. Полипептид PDCT и/или CPT может являться эндогенным для растения или его части, но присутствовать на увеличенном уровне по сравнению с диким типом, или может являться гетерологичным для вида растения. Каждый экзогенный полинуклеотид является функционально связанным с промотором, экспрессируемым в вегетативной части растения, для обеспечения увеличенного уровня или активности PDCT и/или CPT. В предпочтительном варианте осуществления, увеличенные уровень или активность PDCT и/или CPT обеспечивают увеличенную скорость перехода DAG в PC, или PC в DAG, или более предпочтительно, оба из них. Вегетативная часть растения, таким образом, имеет увеличенное количество DAG, доступного для получения TAG посредством активности DGAT. В более предпочтительном варианте осуществления, уровень TAG в вегетативной части растения является увеличенным, по сравнению с соответствующей частью, лишенной экзогенных полинуклеотидов.
В другом аспекте настоящее изобретение относится к растению, предпочтительно, растению вида Sorghum и/или Zea mays, или его части, где растение содержит вегетативную часть растения, имеющую уменьшенный уровень или активность PDCT и/или CPT по сравнению с диким типом. В одном варианте осуществления, растение или его часть содержит мутацию в эндогенном гене(генах), кодирующем фермент(ы), или один или несколько экзогенных полинуклеотидов, каждый из которых кодирует молекулу РНК, уменьшающую экспрессию одного или обоих из эндогенных генов, кодирующих ферменты.
В другом аспекте настоящее изобретение относится к растению, предпочтительно, растению вида Sorghum и/или Zea mays, или его части, где растение содержит вегетативную часть, имеющую увеличенный уровень или активность PLC или PLD, или обоих. В предпочтительном варианте осуществления, растение или его часть содержат один или несколько экзогенных полинуклеотидов, кодирующих полипептид PLC и/или PLD. Полипептид PLC и/или PLD может являться эндогенным для растения или его части, но имеющим увеличенные уровень или активность, по сравнению с диким типом, или может являться гетерологичным для вида растения. Каждый экзогенный полинуклеотид является функционально связанным с промотором, экспрессируемым в вегетативной части растения, для обеспечения увеличенного уровня или активности PLC и/или PLD. В предпочтительном варианте осуществления, увеличенные уровень или активность PLC и/или PLD обеспечивают увеличенную скорость перевода PC в DAG и фосфохолин в случае PLC, или PA и холин в случае PLD. Вегетативная часть, таким образом, имеет увеличенное количество DAG, доступного для получения TAG посредством активности DGAT. В более предпочтительном варианте осуществления, уровень TAG в вегетативной части растения является увеличенным, по сравнению с соответствующей частью, лишенной экзогенных полинуклеотидов, кодирующих PLC и/или PLD.
В другом аспекте настоящее изобретение относится к растению, предпочтительно, к растению вида Sorghum и/или Zea mays, или его части, где растение имеет увеличенные уровень или активность PDAT в вегетативной части, по сравнению с диким типом. В предпочтительном варианте осуществления, вегетативная часть содержит один или несколько экзогенных полинуклеотидов, кодирующих полипептид PDAT. Полипептид PDAT может являться эндогенным для вегетативной части, но имеющим увеличенные уровень или активность, по сравнению с диким типом, или может являться гетерологичным для вида растения. Экзогенный полинуклеотид является функционально связанным с промотором, экспрессируемым в вегетативной части растения, для обеспечения увеличенного уровня или активности PDAT. Является желательным, чтобы увеличенные уровень или активность PDAT обеспечивали увеличенное получение TAG из DAG и PC. В предпочтительном варианте осуществления, увеличенные уровень или активность PDAT присутствуют в комбинации с увеличенными уровнем или активностью PDCT и/или CPT, или с увеличенными уровнем или активностью PLC и/или PLD, как описано выше.
В другом аспекте настоящее изобретение относится к растению, предпочтительно, растению вида Sorghum и/или Zea mays, или его части, где растение имеет увеличенные уровень или активность двух различных DGAT в вегетативной части, по сравнению с диким типом. В предпочтительном варианте осуществления, растение или его часть содержит два экзогенных полинуклеотида, каждый из которых кодирует полипептид DGAT, где полипептиды являются различными. Один из полипептидов DGAT может являться эндогенным для растения или его части, но имеющим увеличенные уровень или активность, по сравнению с диким типом, в то время как другой является гетерологичным, или оба DGAT являются гетерологичными для вида растения. Каждый из экзогенных полинуклеотидов является функционально связанным с промотором, экспрессируемым в вегетативной части, для обеспечения увеличенной экспрессии каждого из DGAT. Является желательным, чтобы увеличенные уровень или активность двух DGAT обеспечивали (i) увеличенную продукцию TAG, полученного из de novo DAG и (ii) увеличенную продукцию TAG, полученного из DAG, полученного из PC (происходящего из PC DAG). В одном варианте осуществления, первый DGAT является более активным по отношению к de novo DAG, чем к происходящему из PC DAG, в то время как второй DGAT является более активным по отношению к происходящему из PC DAG, чем к de novo DAG. Такие различия в активности DGAT могут возникать в результате различной компартментализации или локализации двух DGAT в эндоплазматическом ретикулуме (ER) клетки. В одном варианте осуществления, один из двух DGAT происходит из одноклеточного организма, например, бактериальный DGAT или DGAT одноклеточной водоросли, такой как DGAT Chlamydomonas или его вариант. В предпочтительном варианте осуществления, первый DGAT происходит из вида растения, естественным образом образующего масло, имеющего низкий уровень полиненасыщенных жирных кислот (PUFA), например, менее чем 20% PUFA, в то время как второй DGAT происходит из масличной культуры, образующей относительно более высокий уровень PUFA, например, по меньшей мере 40% PUFA. Как правило, первый DGAT является более активным по отношению к de novo DAG, и второй DGAT является более активным по отношению к происходящему из PC DAG. В предпочтительном варианте осуществления, увеличенная активность двух DGAT присутствует в комбинации с увеличенной активностью PDCT и/или CPT, или с увеличенной активностью PLC и/или PLD, или с увеличенной активностью PDAT, как описано выше.
Комбинации признаков вышеуказанных аспектов явно предусмотрены для растения, части растения и клетки по изобретению, и в способах их получения и использования.
В одном варианте осуществления вышеуказанных аспектов, растение по изобретению является фенотипически нормальным. В одном варианте осуществления, растение по изобретению имеет надземную биомассу, составляющую по меньшей мере 80%, по сравнению с соответствующим растением дикого типа. Предпочтительно, растение имеет высоту растения, составляющую по меньшей мере 80% по сравнению с соответствующим растением дикого типа, и имеет мужскую и женскую фертильность. В одном варианте осуществления, растение представляет собой гибридное растение Zea mays.
В одном варианте осуществления вышеуказанных аспектов, вегетативная часть растения из растения по изобретению имеет общее содержание жирных кислот, отличающееся TTQ между 0,01 и 0,6. В одном варианте осуществления, вегетативная часть растения имеет TTQ между 0,01 и 0,55 или между 0,01 и 0,5, или приблизительно 0,1, или приблизительно 0,2, или приблизительно 0,3, или приблизительно 0,4 или приблизительно 0,5. Предпочтительно, TTQ составляет между 0,60 и 0,84 или между 0,84 и 0,95.
В предпочтительном варианте осуществления вышеуказанных аспектов, клетка или вегетативная часть растения по изобретению содержит один или несколько экзогенных полинуклеотидов или генетических модификаций, каждая из которых, или в комбинации, увеличивает TTQ общего содержания жирных кислот в клетке или вегетативная часть растения, по сравнению с соответствующими клеткой или вегетативной частью растения, лишенной экзогенного полинуклеотида или генетической модификации, где экзогенный полинуклеотид или генетическая модификация обеспечивают (i) a уменьшенное содержание полипептида TAG-липазы, предпочтительно, уменьшенное содержание полипептида SDP1, (ii) уменьшенное содержание полипептида TGD, предпочтительно, уменьшенное содержание полипептида TGD5, (iii) увеличенное содержание полипептида OBC или LDAP, (iv) увеличенное содержание полипептида, увеличивающего экспорт жирных кислот из пластид, предпочтительно, ацил-ACP-тиоэстеразы, (v) уменьшенное содержание полипептида TST, предпочтительно, уменьшенное содержание полипептида TST1, (vi) модифицированный уровень полипептида PDCT, (vii) модифицированный уровень полипептида CPT, (viii) увеличенные уровень или активность полипептида PLC, (ix) увеличенные уровень или активность полипептида PLD, (x) увеличенные уровень или активность полипептида PDAT и (xi) увеличенные уровень или активность двух полипептидов DGAT. Более предпочтительно, TTQ составляет между 0,60 и 0,84 или между 0,84 и 0,95, и/или клетка или вегетативная часть растения имеет содержание TAG приблизительно 6% (масс./масс. сухой массы), предпочтительно, между приблизительно 6% и приблизительно 20%.
В одном варианте осуществления вышеуказанных аспектов, растение или его часть содержит один или оба из
a) первого экзогенного полинуклеотида, кодирующего полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в растении или его части, предпочтительно, полипептид WRI1, и
b) второй экзогенный полинуклеотид, кодирующий полипептид, вовлеченный в биосинтез одного или нескольких неполярных липидов, предпочтительно, DGAT и/или PDAT, и в каждом случае, любое одно или два, или три, или все четыре из
c) генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в катаболизм триацилглицеринов (TAG) в растении или его части, по сравнению с соответствующими растением или его частью, лишенными генетической модификации, предпочтительно, TAG-липазы SDP1,
d) третьего экзогенного полинуклеотида, кодирующего полипептид, который увеличивает экспорт жирных кислот из пластид клетки в растении или его части, по сравнению с соответствующей клеткой, лишенной третьего экзогенного полинуклеотида, предпочтительно, ацил-ACP тиоэстеразу,
e) четвертого экзогенного полинуклеотида, кодирующего второй полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в клетке в растении или его части, предпочтительно, полипептид LEC2, и
f) второй генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в импорт жирных кислот в пластиды клетки, по сравнению с соответствующей клеткой, лишенной второй генетической модификации,
где каждый экзогенный полинуклеотид является функционально связанным с промотором, способным управлять экспрессией полинуклеотида в растении или его части.
В одном варианте осуществления вышеуказанных аспектов, растение или его часть дополнительно содержат одно или оба из
a) пятого экзогенного полинуклеотида, кодирующего полипептид покрытия масляных телец (OBC) или ассоциированный с липидными капельками белок (LDAP), и
b) третьей генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в продукцию диацилглицерина (DAG) в пластидах, по сравнению с соответствующими растением или его частью, лишенными, третьей генетической модификации.
Альтернативно, в следующем варианте осуществления, растение или его часть содержит
a) первый экзогенный полинуклеотид, кодирующий полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в растении или его части, предпочтительно, полипептид WRI1,
b) второй экзогенный полинуклеотид, кодирующий полипептид, вовлеченный в биосинтез одного или нескольких неполярных липидов, предпочтительно, полипептид DGAT и/или полипептид PDAT, и любое одно или два, или все три из
c) генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в катаболизм триацилглицеринов (TAG) в растении или его части, предпочтительно, TAG-липазы SDP1, по сравнению с соответствующими растением или его частью, лишенными генетической модификации,
d) третьего экзогенного полинуклеотида, кодирующего полипептид, который увеличивает экспорт жирных кислот из пластид клетки в растении, по сравнению с соответствующей клеткой, лишенной третьего экзогенного полинуклеотида, предпочтительно, ацил-ACP тиоэстеразу, и
e) четвертого экзогенного полинуклеотида, кодирующего второй полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в клетке в растении или его части, предпочтительно, полипептид LEC2,
где каждый экзогенный полинуклеотид является функционально связанным с промотором, способным управлять экспрессией полинуклеотида в растении или его части.
В одном варианте осуществления вышеуказанного аспекта, растение или его часть дополнительно содержат одно или несколько, или все из
a) пятого экзогенного полинуклеотида, кодирующего полипептид покрытия масляных телец (OBC),
b) второй генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в импорт жирных кислот в пластиды растения, по сравнению с соответствующим растением, лишенным второй генетической модификации, и
c) третьей генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в продукцию диацилглицерина (DAG) в пластиде, по сравнению с соответствующим растением, лишенным третьей генетической модификации.
В следующем варианте осуществления вышеуказанных аспектов, часть растения представляет собой вегетативную часть растения, и один или несколько, или все из промоторов экспрессируются на более высоком уровне в вегетативной части растения, по сравнению с семенем растения. Например, предпочтительный промотор представляет собой промотор гена убиквитина или промотор SSU. Альтернативно, один или несколько, или все из промоторов являются отличными от промотора SSU.
В следующем варианте осуществления, растение или его часть дополнительно отличается одним или несколькими признаками, как описано в контексте клетки по изобретению, или способов из вышеуказанных аспектов.
В одном варианте осуществления вышеуказанных аспектов, растение вида Sorghum и/или Zea mays по изобретению, или растение или его часть, используемые в способе по изобретению или иначе описанные в настоящем описании, выращивали с фотопериодом по меньшей мере 13 часов в сутки в течение периода по меньшей мере 1 недели или по меньшей мере 2 недель, или по меньшей мере 3 недель, или по меньшей мере 4 недель, предпочтительно, вплоть до сбора растения для получения вегетативных частей из растения. В таких условиях, надземная биомасса растения, предпочтительно, составляет по меньшей мере 80% по сравнению с соответствующим растением дикого типа. Семена растения можно собирать из растения после роста в таких условиях.
В другом варианте осуществления вышеуказанных аспектов, растение вида Sorghum и/или Zea mays по изобретению, или растение или его часть, используемые в способе по изобретению или иначе описанные в настоящем описании, выращивали/выращивают при концентрации CO2 по меньшей мере 400 м.д.
В следующем варианте осуществления, a растение вида Sorghum и/или Zea mays по изобретению, или растение или его часть, используемые в способе по изобретению или иначе описанные в настоящем описании, содержат один или несколько экзогенных полинуклеотидов, кодирующих один или несколько белков, увеличивающих общее содержание белка в вегетативной части растения.
В другом аспекте настоящее изобретение относится к популяции из по меньшей мере приблизительно 1000 растений, где каждое представляет собой растение по изобретению, растущее в поле, или к коллекции из по меньшей мере приблизительно 1000 вегетативных частей растений, где каждая представляет собой вегетативную часть растения по изобретению, где вегетативные частях растений собраны из растений, растущих в поле. Предпочтительно, растения выращивали в условиях фотопериода и/или CO2, описанных выше.
В другом аспекте настоящее изобретение относится к семени растения или к семени, полученному из растения по изобретению, или к семени, из которого при посеве получают растения по изобретению. Альтернативно, семя может являться обработанным таким образом, что оно более неспособно прорастать, и/или является измельченным, размолотым, шлифованным, расщепленным или подвергнутым термообработке.
В следующем аспекте, настоящее изобретение относится к способу идентификации, отбора и/или получения растения или его части по изобретению, предпочтительно, растения вида Sorghum и/или Zea mays, или его части, с желательным фенотипом, включающему
i) получение множества растений-кандидатов или их частей, каждое из которых содержит один или оба из
a) первого экзогенного полинуклеотида, кодирующего полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в растении или его части, предпочтительно, полипептид WRI1, и
b) второго экзогенного полинуклеотида, кодирующего полипептид, вовлеченный в биосинтез одного или нескольких неполярных липидов, предпочтительно, полипептид DGAT и/или полипептид PDAT, и в каждом случае, любое одно или два, или три, или все четыре из
c) генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в катаболизм триацилглицеринов (TAG) в растении или его части, по сравнению с соответствующими растением или его частью, лишенными генетической модификации, предпочтительно, TAG-липазы SDP1,
d) третьего экзогенного полинуклеотида, кодирующего полипептид, который увеличивает экспорт жирных кислот из пластид клетки в растении или его части, по сравнению с соответствующей клеткой, лишенной третьего экзогенного полинуклеотида, предпочтительно, ацил-ACP-тиоэстеразу,
e) четвертого экзогенного полинуклеотида, кодирующего второй полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в клетке в растении или его части, предпочтительно, полипептид LEC2, и
f) второй генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в импорт жирных кислот в пластиды клетки, по сравнению с соответствующей клеткой, лишенной второй генетической модификации,
где каждый экзогенный полинуклеотид является функционально связанным с промотором, способным управлять экспрессией полинуклеотида в растении;
ii) анализ липидов во множестве частей, или по меньшей мере, в части каждого растения во множестве растений-кандидатов, со стадии i), и
iii) идентификацию, отбор и/или получение растения, или его части, содержащих вегетативную часть растения, общее содержание жирных кислот (TFA) в которой включает жирные кислоты, этерифицированные в форме триацилглицеринов (TAG), и жирные кислоты в форме липидов, отличных от TAG, и содержащих вегетативную часть растения, имеющую содержание TFA приблизительно 5% (масс./масс. сухой массы), предпочтительно, между приблизительно 6% и приблизительно 20%.
В одном варианте осуществления, отбирают растение или его часть, содержащие вегетативную часть растения, общее содержание жирных кислот в которой отличается наличием TTQ между 0,01 и 0,6. В одном варианте осуществления, отбирают растение или его часть, где растение содержит вегетативную часть растения, имеющую TTQ между 0,01 и 0,55 или между 0,01 и 0,5, или bout 0,1, или приблизительно 0,2, или приблизительно 0,3, или приблизительно 0,4 или приблизительно 0,5. В предпочтительном варианте осуществления, TTQ составляет между 0,60 и 0,84 или между 0,84 и 0,95.
В другом аспекте настоящее изобретение относится к способу идентификации, отбора и/или получения растения, предпочтительно, растения вида Sorghum и/или Zea mays, или его части, с желательным фенотипом, включающему
i) получение множества растений-кандидатов или их частей, каждое из которых содержит один или оба из
a) первого экзогенного полинуклеотида, кодирующего полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в растении или его части, предпочтительно, полипептид WRI1,
b) второго экзогенного полинуклеотида, кодирующего полипептид, вовлеченный в биосинтез одного или нескольких неполярных липидов, предпочтительно, полипептид DGAT и/или полипептид PDAT, и любое одно или два, или все три из
c) генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в катаболизм триацилглицеринов (TAG) в растении или его части, предпочтительно, TAG-липазы SDP1, по сравнению с соответствующими растением или его частью, лишенными генетической модификации,
d) третьего экзогенного полинуклеотида, кодирующего полипептид, который увеличивает экспорт жирных кислот из пластид клетки в растении или его части, по сравнению с соответствующей клеткой, лишенной третьего экзогенного полинуклеотида, предпочтительно, ацил-ACP-тиоэстеразу, и
e) четвертого экзогенного полинуклеотида, кодирующего второй полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в клетке в растении или его части, предпочтительно, полипептид LEC2,
где каждый экзогенный полинуклеотид является функционально связанным с промотором, способным управлять экспрессией полинуклеотида в растении или его части;
ii) анализ липидов во множестве частей, или по меньшей мере, в части каждого растения во множестве растений-кандидатов, со стадии i),
iii) идентификацию, отбор и/или получение растения, или его части, где растение содержит вегетативную часть растения, общее содержание жирных кислот в которой включает жирные кислоты, этерифицированные в форме триацилглицеринов (TAG), и жирные кислоты в форме липидов, отличных от TAG, и содержащих вегетативную часть растения, имеющую общее содержание TAG приблизительно 6% (масс./масс. сухой массы), предпочтительно, между приблизительно 6% и приблизительно 20%, и предпочтительно, имеющую соотношение жирных кислот, этерифицированных в форме TAG, к жирным кислотам в форме липидов, отличных от TAG, составляющее между 20:1 и 1,5:1 или между 5:1 и 2:1.
В другом аспекте настоящее изобретение относится к способу идентификации, отбора и/или получения растения, предпочтительно, растения вида Sorghum и/или Zea mays, или его части, имеющих увеличенное TTQ в общем содержании жирных кислот, включающему
i) получение множества растений-кандидатов или их частей, каждое из которых содержит одну или несколько генетических модификаций, обеспечивающих (a) уменьшенное содержание или активность полипептида TAG-липазы, предпочтительно, уменьшенное содержание или активность TAG-липазы SDP1, (b) уменьшенное содержание или активность полипептида TGD, предпочтительно, уменьшенное содержание или активность полипептида TGD5, (c) увеличенное содержание полипептида OBC или LDAP, (d) увеличенное содержание или активность полипептида, который увеличивает экспорт жирных кислот из пластид, предпочтительно, ацил-ACP-тиоэстеразы, (e) уменьшенное содержание или активность полипептида TST, предпочтительно, уменьшенное содержание или активность полипептида TST1, (f) модифицированный уровень или активность полипептида PDCT, (g) модифицированный уровень или активность полипептида CPT, (h) увеличенное содержание или активность полипептида PLC, (i) увеличенное содержание или активность полипептида PLD, (j) увеличенное содержание или активность полипептида PDAT и (k) увеличенное содержание или активность двух полипептидов DGAT,
ii) анализ липидов во множестве растений, или по меньшей мере, в части каждого растения во множестве растений-кандидатов, со стадии i),
iii) идентификацию, отбор и/или получение растения, или его части, имеющих увеличенное TTQ в общем содержании жирных кислот, по сравнению с соответствующими растением или частью растения, лишенными генетических модификаций, или по сравнению с другим растением или частью растения из множества растений-кандидатов или их частей.
В одном варианте осуществления вышеуказанного аспекта, увеличенное TTQ увеличено по меньшей мере на 0,05, предпочтительно, между 0,5 и 0,80. Каждая из одной или нескольких генетических модификаций, при экспрессии в растениях-кандидатах или их частях, приводит к уменьшенным содержанию или активности полипептида в соответствии с (a), (b) или (e), к увеличенным содержанию или активности в соответствии с (c), (d), (h), (i), (j) или (k), и либо к увеличенным, либо к уменьшенным содержанию и/или активности для (f) или (g). В случае уменьшенных содержания или активности полипептида, каждая генетическая модификация независимо представляет собой мутацию эндогенного гена, кодирующего полипептид, которая частично или полностью инактивирует ген, такую как точечная мутация, инсерция, или предпочтительно, делеция, или генетическая модификация включает интеграцию в геном экзогенного полинуклеотида, который кодирует молекулу РНК, ингибирующую экспрессию эндогенного гена, где экзогенный полинуклеотид является функционально связанным с промотором, способным управлять экспрессией полинуклеотида в растении или его части.
В одном варианте осуществления, стадия (ii) способа из вышеуказанных аспектов включает экстракцию липида из растений-кандидатов или их частей, и одно или несколько из (a) отделения TAG от не относящегося к TAG липида в растенииях или их частях, (b) определение относительных количеств TAG и не относящегося к TAG липида в экстрагированном липиде. В одном варианте осуществления, способ включает стадию расчета TTQ для растений-кандидатов или их частей, после стадии ii). Идентифицированное или отобранное растение может быть идентифицировано или отобрано на основании его TTQ и/или его содержания TAG или TFA. В предпочтительном варианте осуществления, идентифицированное отобранное растение содержит вегетативную часть растения, имеющую TTQ, составляетющее между 0,60 и 0,84 или между 0,84 и 0,95, и/или вегетативная часть растения имеет содержание TAG приблизительно 6% (масс./масс. сухой массы), предпочтительно, между приблизительно 6% и приблизительно 20%.
В одном варианте осуществления способа из вышеуказанных аспектов, способ дополнительно включает стадию размножения растения или его части по изобретению для получения потомства растений или их частей, например, из семян или вегетативных частей из растения, или скрещивания растения с растением другого генетического состава для введения генетической модификации(модификаций) в другой генетический фон. Изобретение явно включает растения-потомки и их части, содержащие генетическую модификацию(модификации) и имеющие увеличенное TTQ в общем содержании жирных кислот.
В более предпочтительном варианте осуществления способа из вышеуказанных аспектов, каждое из множества растений-кандидатов или их частей содержит первый экзогенный полинуклеотид, кодирующий полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в растении или его части, предпочтительно, полипептид WRI1, и второй экзогенный полинуклеотид, кодирующий полипептид, вовлеченный в биосинтез одного или нескольких неполярных липидов, предпочтительно, полипептид DGAT и/или полипептид PDAT. В этом предпочтительном варианте осуществления, генетическая модификация(модификации), приводящие к уменьшенным, увеличенным или модифицированным содержанию или активности полипептида в соответствии с (a) - (k), являются дополнительными к первому и второму экзогенным полинуклеотидам, и увеличивают TTQ по сравнению с соответствующим растением или вегетативной частью, имеющим первый и второй экзогенный полинуклеотиды, но лишенным генетической модификации(модификаций).
В одном варианте осуществления способа из вышеуказанных аспектов, растение или его часть, идентифицированные, отобранные и/или полученные, дополнительно отличаются одним или несколькими признаками, как определено в контексте растения по изобретению, растения вида Sorghum и/или Zea mays по изобретению, или способов из вышеуказанных аспектов.
Способ идентификации, отбора и/или получения растения по изобретению можно также использовать для идентификации, отбора и/или получения растения, имеющего увеличенное TTQ или содержание TAG в стебле растения, которое, предпочтительно, сопровождается увеличенным TTQ или содержанием TAG в листьях растения, хотя TTQ и содержание TAG в листьях растения могут являться совсем не увеличенными или не увеличенными настолько, как в стебле.
В другом аспекте настоящее изобретение относится к способу получения клетки или растения по изобретению, предпочтительно, клетки или растения вида Sorghum или Zea mays, включающему стадии введения в клетку или растение, предпочтительно, в клетку или растение вида Sorghum или Zea mays, по меньшей мере одного экзогенного полинуклеотида и/или по меньшей мере одной генетической модификации, как определено выше.
В одном варианте осуществления, способ включает одну или несколько, или все стадии из
i) экспрессии экзогенного полинуклеотида(полинуклеотидов) и/или генетических модификаций в клетке или растении, или в являющихся их потомками клетке или растении,
ii) анализа содержания липидов в клетке или растении, или в клетке-потомке или растении-потомке, и
iii) отбора или идентификации клетки или растения по изобретению.
Полученная клетка может находиться в растении Sorghum или Zea mays, или предпочтительно, в его вегетативной части.
В одном варианте осуществления, экзогенный полинуклеотид(ы) и/или генетические модификации обеспечивают модифицированный признак, включающий уменьшенное, увеличенное или модифицированное содержание полипептида, в соответствии с (a) - (k) выше. В одном варианте осуществления, способ включает стадию расчета TTQ для растений-кандидатов или их частей, после стадии ii). В предпочтительном варианте осуществления, клетка или растение отобраны или идентифицированы на основании их TTQ и/или их содержания TAG, более предпочтительно, TTQ, составляющего между 0,60 и 0,84 или между 0,84 и 0,95, и/или вегетативная часть растения имеет содержание TAG приблизительно 6% (масс./масс. сухой массы), предпочтительно, между приблизительно 6% и приблизительно 20%.
В другом аспекте настоящее изобретение относится к способу получения растения, предпочтительно, растения вида Sorghum и/или Zea mays, в геном которого интегрирован набор экзогенных полинуклеотидов и/или генетических модификаций определенных в настоящем описании, включающему стадии
i) скрещивания двух материнских растений, где одно растение содержит по меньшей мере один из экзогенных полинуклеотидов и/или по меньшей мере одну из генетических модификаций, как определено выше, и другое растение содержит по меньшей мере один из экзогенных полинуклеотидов и/или по меньшей мере одну из генетических модификаций, как определено выше, и при этом два материнских растения содержат набор экзогенных полинуклеотидов и/или генетических модификаций, как определено выше,
ii) скрининга одного или нескольких растений-потомков скрещивания на предмет присутствия или отсутствия набора экзогенных полинуклеотидов и/или генетических модификаций, как определено выше, и
iii) отбора растения-потомка, содержащего набор экзогенных полинуклеотидов и/или генетических модификаций, как определено выше,
с получением таким образом растения.
В одном варианте осуществления, полученное растение или его часть дополнительно отличается одним или несколькими признаками, как описано в контексте клетки или растения по изобретению, предпочтительно, клетки или растения вида Sorghum или Zea mays, или способов из вышеуказанных аспектов.
В другом аспекте, настоящее изобретение относится к способу получения нефтепродукта, включающему стадии
(i) обработки в реакторе композиции, содержащей
(a) вегетативные части растений, предпочтительно, вегетативные части растений вида Sorghum или Zea mays, общее содержание жирных кислот (TFA) в которых включает жирные кислоты, этерифицированные в форме триацилглицеринов (TAG), и жирные кислоты в форме липидов, отличных от TAG, где вегетативная часть растения имеет содержание TFA приблизительно 5% (масс./масс. сухой массы), предпочтительно, по меньшей мере 10%,
(b) растворитель, который содержит воду, спирт или и то, и другое, и
(c) необязательно, катализатор,
где обработка включает нагревание композиции при температуре от приблизительно 50°C до приблизительно 450°C и давлении от 5 до 350 бар на протяжении периода от 1 до 120 минут, в окислительной, восстановительной или инертной среде,
(ii) выделения нефтепродукта из реактора с выходом по меньшей мере 35% масс., в пересчете на сухую массу вегетативных частей растений,
с получением таким образом нефтепродукта.
В одном варианте осуществления, вегетативные части растений имеют TTQ между 0,01 и 0,6. В одном варианте осуществления, вегетативные части растений имеют TTQ между 0,01 и 0,55, или между 0,01 и 0,5, или приблизительно 0,1, или приблизительно 0,2 или приблизительно 0,3, или приблизительно 0,4, или приблизительно 0,5. Предпочтительно, TTQ составляет между 0,60 и 0,84 или между 0,84 и 0,95.
В другом аспекте настоящее изобретение относится к способу получения нефтепродукта, включающему стадии
(i) обработки в реакторе композиции, содержащей
(a) вегетативные части растений, предпочтительно, вегетативные части растений вида Sorghum или Zea mays, общее содержание жирных кислот в которых включает жирные кислоты, этерифицированные в форме триацилглицеринов (TAG), и жирные кислоты в форме липидов, отличных от TAG, где вегетативная часть растения имеет общее содержание TAG приблизительно 6% (масс./масс. сухой массы) и предпочтительно, имеет соотношение жирных кислот, этерифицированных в форме TAG, к жирным кислотам в форме липидов, отличных от TAG, составляющее между 20:1 и 1,5:1 или между 5:1 и 2:1,
(b) растворитель, который содержит воду, спирт или и то, и другое, и
(c) необязательно, катализатор,
где обработка включает нагревание композиции при температуре между приблизительно 50°C и приблизительно 450°C и давлении от 5 до 350 бар на протяжении периода от 1 до 120 минут в окислительной, восстановительной или инертной среде,
(ii) выделения нефтепродукта из реактора с выходом по меньшей мере 35% масс., в пересчете на сухую массу вегетативных частей растений,
с получением таким образом нефтепродукта.
В одном варианте осуществления двух вышеуказанных аспектов, используют одно или несколько, или все, из следующего:
(i) вегетативные части растений имеют сухую массу по меньшей мере 1 кг,
(ii) вегетативные части растений имеют содержание TFA и/или общее содержание неполярных липидов по меньшей мере 10%, по меньшей мере 15%, по меньшей мере 20%, приблизительно 25%, приблизительно 30%, приблизительно 35%, или между 30% и 75% в пересчете на сухую массу,
(iii) концентрация твердых веществ в композиции составляет между 5% и 90%,
(iv) катализатор содержит NaOH или KOH, или оба, предпочтительно, в концентрации 0,1-2 М,
(v) время обработки составляет между 1 и 60 минут, предпочтительно, между 10 и 60 минут, более предпочтительно, между 15 и 30 минут,
(vi) если растворитель представляет собой воду, способ обеспечивает выход нефтепродукта между минимум 36%, 37%, 38%, 39% или 40% и максимум 55% или 60% масс., в пересчете на сухую массу вегетативных частей растений,
(vii) если растворитель содержит спирт, способ обеспечивает выход нефтепродукта между минимум 36%, 37%, 38%, 39% или 40% и максимум 65% или 70% масс., в пересчете на сухую массу вегетативных частей растений,
(viii) если растворитель содержит приблизительно 80% воды, нефтепродукт содержит приблизительно 30% C13-C22 углеводородных соединений,
(ix) если растворитель содержит приблизительно 50% метанола, нефтепродукт содержит приблизительно 50% метиловых сложных эфиров жирных кислот (FAME),
(x) содержание воды в выделенном нефтепродукте составляет менее чем приблизительно 15% по массе,
(xi) выход нефтепродукта по меньшей мере на 2% масс. больше, чем выход соответствующего способа с использованием соответствующих вегетативных частей растений, в которых содержание неполярных липидов составляет менее чем 2% в пересчете на сухую массу, и
(xii) вегетативные части растений на стадии (i) (a) физически обрабатывают посредством одного или нескольких из сушки, рубки, шинковки, помола, вальцевания, прессования, дробления или перетирания.
В следующем варианте осуществления двух вышеуказанных аспектов, способ дополнительно включает одно или несколько из:
(i) гидродезоксигенации выделенного нефтепродукта,
(ii) обработки выделенного нефтепродукта водородом, с целью уменьшения уровней кетонов или сахаров в нефтепродукте,
(iii) получения синтетического газа из выделенного нефтепродукта, и
(iv) фракционирования выделенного нефтепродукта, с целью получения одного или нескольких из мазута, дизельного топлива, керосина или бензина.
В следующем варианте осуществления двух вышеуказанных аспектов, вегетативные части растений включают листья растения, стебли или и то, и другое.
В одном варианте осуществления двух вышеуказанных аспектов, обработанные вегетативные части растений дополнительно отличаются одним или несколькими признаками, как определено в контексте частей или клеток растения вида Sorghum и/или Zea mays по изобретению.
В другом аспекте настоящее изобретение относится к способу производства промышленного продукта, включающему стадии:
i) получения клетки по изобретению, предпочтительно, клетки, растения или его части вида Sorghum или Zea mays по изобретению, предпочтительно, растения вида Sorghum и/или Zea mays или его части, или семени по изобретению, и
ii) либо
a) перевода по меньшей мере части липида в клетке, растении или его части, или семени со стадии i) в промышленный продукт, с применением нагревания, химических или ферментных средств или любой их комбинации, к липиду in situ в клетке, растении или его части, или семени, или
b) физической обработки клетки, растения или его части, или семени со стадии i), и одновременного или последующего перевода по меньшей мере части липида в обрабатываемой клетке, растении или его части, или семени в промышленный продукт, с применением нагревания, химических или ферментных средств или любой их комбинации к липиду в обрабатываемой клетке, растении или его части, или семени, и
iii) выделения промышленного продукта,
с получением таким образом промышленного продукта.
В одном варианте осуществления, часть растения представляет собой вегетативную часть растения по изобретению.
В одном варианте осуществления, стадия физической обработки клетки, растения или его части, или семени, включает одно или несколько из вальцевания, прессования, дробления или перетирания клетки, растения или его части, или семени. Промышленный продукт является таким, как описано в настоящем описании.
В следующем варианте осуществления, способ дополнительно включает стадии:
(a) экстракции по меньшей мере части неполярного липида, содержащегося в клетке, растении или его части, или семени, в форме неполярного липида, и
(b) выделения экстрагированного неполярного липида,
где стадии (a) и (b) выполняют до стадии перевода по меньшей мере части липида в клетке, растении или его части, или семени, в промышленный продукт.
Экстрагированный неполярный липид предпочтительно, содержит триацилглицерины, где триацилглицерины составляют по меньшей мере 90%, более предпочтительно, по меньшей мере 95%, экстрагированного липида.
В другом аспекте настоящее изобретение относится к способу получения экстрагированного липида, включающему стадии:
i) получения клетки по изобретению, предпочтительно, клетки, растения или его части вида Sorghum или Zea mays по изобретению, предпочтительно, растения вида Sorghum и/или Zea mays или его части, или семени по изобретению,
ii) экстракции липида из клетки, растения или его части, или семени, и
iii) выделения экстрагированного липида,
с получением таким образом экстрагированного липида.
В одном варианте осуществления, стадия экстракции включает одно или несколько из сушки, вальцевания, прессования, дробления или перетирания растения или его части, или семени, и/или очистки экстрагированного липида или масла из семян. В одном варианте осуществления, органический растворитель используют в способе в процессе экстракции, для экстракции масла.
В одном варианте осуществления, способ включает выделение экстрагированного липида посредством сбора его в емкость и/или одного или нескольких из дегуммации, дезодорирования, обесцвечивания, сушки, фракционирования экстрагированного липида, удаления по меньшей мере некоторых восков и/или сложных эфиров восков из экстрагированного липида, или анализа состава жирных кислот экстрагированного липида экстрагированного липида.
В одном варианте осуществления, объем экстрагированного липида или масла составляет по меньшей мере 1 литр.
В следующем варианте осуществления, используют один или несколько, или все из следующих признаков:
(i) экстрагированный липид или масло содержит триацилглицерины, где триацилглицерины составляют по меньшей мере 90%, предпочтительно, по меньшей мере 95% или по меньшей мере 96%, экстрагированного липида или масла,
(ii) экстрагированный липид или масло содержит свободные стеролы, стероильные сложные эфиры, стероилгликозиды, воски или сложные эфиры восков или любую их комбинацию, и
(iii) общее содержание и/или состав стеролов в экстрагированном липиде или масле значительно отличается от содержания и/или состава стеролов в экстрагированном липиде или масле, полученном из соответствующего растения или его части, или семени.
В следующем варианте осуществления, способ дополнительно включает перевод экстрагированного липида или масла в промышленный продукт.
В следующем варианте осуществления, промышленный продукт является углеводородным продуктом, таким как сложные эфиры жирных кислот, предпочтительно, метиловые сложные эфиры жирных кислот и/или этиловые сложные эфиры жирных кислот, алкан, такой как метан, этан или длинноцепочечный алкан, смесь алканов с более длинной цепью, алкен, биотопливо, газообразный монооксид углерода и/или водород, биоспирт, такой как этанол, пропанол, или бутанол, биоуголь, или комбинация монооксида углерода, водорода и биоугля.
В следующем варианте осуществления, часть растения представляет собой воздушную часть растения или зеленую часть растения, предпочтительно, вегетативную часть растения, такую как лист или стебель растения.
В другом варианте осуществления, стадия получения растения или его части включает стадию сбора урожая растения или его части с помощью механического сборщика.
В другом варианте осуществления, уровень липида в растении или его части, или семени, и/или в экстрагированном липиде или масле, поддается определению посредством анализа с использованием газовой хроматографии метиловых сложных эфиров жирных кислот, полученных из экстрагированного липида или масла.
В другом варианте осуществления, часть растения представляет собой вегетативную часть растения, имеющую общее содержание TAG по меньшей мере приблизительно 11%, по меньшей мере приблизительно 12%, по меньшей мере приблизительно 15%, по меньшей мере приблизительно 20%, по меньшей мере приблизительно 25%, по меньшей мере приблизительно 30%, по меньшей мере приблизительно 35%, по меньшей мере приблизительно 40%, по меньшей мере приблизительно 45%, по меньшей мере приблизительно 50%, по меньшей мере приблизительно 55%, по меньшей мере приблизительно 60%, по меньшей мере приблизительно 65%, по меньшей мере приблизительно 70%, между 8% и 75%, между 10% и 75%, между 11% и 75%, между приблизительно 15% и 75%, между приблизительно 20% и 75%, между приблизительно 30% и 75%, между приблизительно 40% и 75%, между приблизительно 50% и 75%, между приблизительно 60% и 75%, или между приблизительно 25% и 50% (масс./масс. сухой массы).
В одном варианте осуществления вышеуказанных аспектов, используемые клетки, растения или их части, или семена, дополнительно отличаются одним или несколькими признаками, как определено в контексте частей или клеток растений по изобретению, предпочтительно, частей или клеток растения вида Sorghum и/или Zea mays.
В другом аспекте настоящее изобретение относится к способу получения семени, включающему:
i) выращивание растения по изобретению, и
ii) сбор урожая семени с растения.
В одном варианте осуществления, способ включает выращивание популяции из по меньшей мере приблизительно 1500, по меньшей мере приблизительно 3000 или по меньшей мере приблизительно 5000 растений, каждое из которых представляет собой растение по изобретению, и сбор урожая семени с популяции растений.
В другом аспекте настоящее изобретение относится к выделенному или экстрагированному липиду, или растворимому белку, который можно получать из клетки по изобретению, предпочтительно, клетки, растения или его части вида Sorghum или Zea mays по изобретению, предпочтительно, растения вида Sorghum и/или Zea mays или его части, семени по изобретению, или который можно получать посредством способа по изобретению.
В другом аспекте настоящее изобретение относится к промышленному продукту, полученному с использованием способа по изобретению, который является углеводородным продуктом, таким как эфиры жирных кислот, предпочтительно, метиловые сложные эфиры жирных кислот и/или этиловые сложные эфиры жирных кислот, алкан, такой как метан, этан или длинноцепочечный алкан, смесь алканов с более длинной цепью, алкен, биотопливо, газообразный монооксид углерода и/или водород, биоспирт, такой как этанол, пропанол, или бутанол, биоуголь или комбинация монооксида углерода, водорода и биоугля.
В другом аспекте, настоящее изобретение относится к применению клетки по изобретению, предпочтительно, клетки, растения или его части вида Sorghum или Zea mays по изобретению, предпочтительно, растения вида Sorghum и/или Zea mays, или его части, семени по изобретению, или выделенного или экстрагированного липида по изобретению для производства промышленного продукта. Примеры промышленных продуктов по изобретению включают примеры, описанные в предшествующем аспекте.
В другом аспекте настоящее изобретение относится к способу получения топлива, включающему:
i) проведение реакции липида по изобретению со спиртом, необязательно в присутствии катализатора, с получением алкиловых сложных эфиров, и
ii) необязательно, смешивание алкиловых сложных эфиров с топливом на основе нефти.
В другом аспекте настоящее изобретение относится к способу получения синтетического дизельного топлива, включающему:
i) перевод липида в клетке по изобретению, предпочтительно, в клетке, растении или его части вида Sorghum или Zea mays по изобретению, предпочтительно, растении вида Sorghum и/или Zea mays, или его части, или семени по изобретению, в бионефть с использованием способа, включающего пиролиз или гидротермическую обработку, или в синтетический газ посредством газификации, и
ii) перевод бионефти в синтетическое дизельное топливо с использованием способа, включающего фракционирование, предпочтительно, с отбором углеводородных соединений, которые конденсируются при температуре между приблизительно 150°C и приблизительно 200°C или между приблизительно 200°C и приблизительно 300°C, или перевод синтетического газа в биотопливо с использованием металлического катализатора или микробного катализатора.
В другом аспекте настоящее изобретение относится к способу получения биотоплива, включающему перевод липида в клетке по изобретению, предпочтительно, в клетке, растении или его части вида Sorghum или Zea mays по изобретению, предпочтительно, в растении вида Sorghum и/или Zea mays, или его части, или семени по изобретению, в бионефть посредством пиролиза, в биоспирт посредством ферментации или в биогаз посредством газификации или анаэробного расщепления.
В одном варианте осуществления, часть представляет собой вегетативную часть растения.
Авторы настоящего изобретения показали также значительные модификации признаков трансгенных растений или их частей, таких как вегетативные части, посредством манипуляции с липидными путями.
Таким образом, в другом аспекте настоящее изобретение относится к трансгенному растению или его части, предпочтительно, растению вида Sorghum и/или Zea mays или его части, содержащим
a) первый экзогенный полинуклеотид, кодирующий полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в растении или его части, предпочтительно, полипептид WRI1,
b) второй экзогенный полинуклеотид, кодирующий полипептид, вовлеченный в биосинтез одного или нескольких неполярных липидов, предпочтительно, полипептид DGAT и/или полипептид PDAT,
c) увеличенное содержание триацилглицерина (TAG) в части или по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью,
и один или несколько, или все из следующих фенотипов;
d) увеличенное содержание растворимого белка в части или по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью,
e) увеличенное содержание азота в части или по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью,
f) уменьшенное соотношение углерод:азот в части или по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью,
g) увеличенная экспрессия фотосинтетического гена в части или по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью,
h) увеличенная способность к фотосинтезу в части или по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью,
i) уменьшенное общее содержание пищевых волокон (TDF) в части или по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью,
j) увеличенное содержание углерода в части или по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью,
k) увеличенное содержание энергии в части или по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью, и
l) увеличенное TTQ по сравнению с соответствующим растением дикого типа или его частью,
где каждый экзогенный полинуклеотид является функционально связанным с промотором, способным управлять экспрессией полинуклеотида в растении или его части.
В одном варианте осуществления, растение или его часть происходит из предкового трансгенного растения, содержащего первый и второй экзогенные полинуклеотиды, где предковое трансгенное растение выбрано из множества трансгенных растений-кандидатов, где каждое содержит первый и второй экзогенные полинуклеотиды, на основании того, что предковое трансгенное растение содержит один или несколько, или все из следующих фенотипов;
a) увеличенного содержания растворимого белка по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью,
b) увеличенного содержания азота по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью,
c) уменьшенного соотношения углерод:азот по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью,
d) увеличенной экспрессии фотосинтетического гена по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью,
e) увеличенной способности к фотосинтезу по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью,
f) уменьшенного общего содержания пищевых волокон (TDF) по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью,
g) увеличенного содержания углерода по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью, и
h) увеличенного содержания энергии по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью.
В другом аспекте настоящее изобретение относится к трансгенному растению или его части, предпочтительно, растению вида Sorghum и/или Zea mays или его части, содержащим
a) первый экзогенный полинуклеотид, кодирующий полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в растении или его части, предпочтительно, полипептид WRI,
b) второй экзогенный полинуклеотид, кодирующий полипептид, вовлеченный в биосинтез одного или нескольких неполярных липидов, предпочтительно, полипептид DGAT и/или полипептид PDAT,
c) увеличенное содержание триацилглицерина (TAG) в части или по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью,
где каждый экзогенный полинуклеотид является функционально связанным с промотором, способным управлять экспрессией полинуклеотида в растении или его части, и где трансгенное растение происходит из предкового трансгенного растения, содержащего первый и второй экзогенные полинуклеотиды, где предковое трансгенное растение выбрано из множества трансгенных растений-кандидатов, где каждое содержит первый и второй экзогенные полинуклеотиды на основании того, что предковое трансгенное растение содержит один или несколько, или все из следующих фенотипов;
i) увеличенного содержания растворимого белка по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью,
ii) увеличенного содержания азота по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью,
iii) уменьшенное соотношение углерод:азот по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью,
iv) увеличенной экспрессии фотосинтетического гена по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью,
v) увеличенной способности к фотосинтезу по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью,
vi) уменьшенного общего содержания пищевых волокон (TDF) по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью,
vii) увеличенного содержания углерода по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью,
viii) увеличенного содержания энергии по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью, и
ix) увеличенного TTQ и/или увеличенного содержания TAG, по сравнению с соответствующим растением дикого типа или его частью.
В одном варианте осуществления двух вышеуказанных аспектов, растение или его часть имеет одно или несколько, или все из;
i) увеличенного содержания растворимого белка по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью,
ii) увеличенного содержания азота по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью,
iii) уменьшенное соотношение углерод:азот по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью.
В одном варианте осуществления, растение или его часть имеет одно или несколько, или все из;
i) растение или его часть имеет увеличенное содержание растворимого белка в части или по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью, по меньшей мере приблизительно на 10%, по меньшей мере приблизительно на 25%, по меньшей мере приблизительно на 50%, по меньшей мере приблизительно на 75%, по меньшей мере приблизительно на 100%, между приблизительно 10% и приблизительно 200%, между приблизительно 50% и приблизительно 150%, или между приблизительно 50% и приблизительно 125%,
ii) растение или его часть имеет увеличенное содержание азота в части или по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью по меньшей мере приблизительно на 10%, по меньшей мере приблизительно на 25%, по меньшей мере приблизительно на 50%, по меньшей мере приблизительно на 75%, по меньшей мере приблизительно на 100%, между приблизительно 10% и приблизительно 200%, между приблизительно 50% и приблизительно 150% или между приблизительно 50% и приблизительно 125%,
iii) часть представляет собой лист, имеющий увеличенное содержание растворимого белка, по сравнению с соответствующим листом дикого типа, по меньшей мере приблизительно на 10%, по меньшей мере приблизительно на 25%, по меньшей мере приблизительно на 50%, по меньшей мере приблизительно на 75%, по меньшей мере приблизительно на 100%, между приблизительно 10% и приблизительно 200%, между приблизительно 50% и приблизительно 150%, или между приблизительно 50% и приблизительно 125%,
iv) часть представляет собой лист, имеющий увеличенное содержание азота по сравнению с соответствующим листом дикого типа, по меньшей мере приблизительно на 10%, по меньшей мере приблизительно на 25%, по меньшей мере приблизительно на 50%, по меньшей мере приблизительно на 75%, по меньшей мере приблизительно на 100%, между приблизительно 10% и приблизительно 200%, между приблизительно 50% и приблизительно 150%, или между приблизительно 50% и приблизительно 125%,
v) растение или его часть имеет уменьшенное соотношение углерод:азот в части или по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью of по меньшей мере приблизительно на 10%, по меньшей мере приблизительно на 25%, по меньшей мере приблизительно 40%, между приблизительно 10% и приблизительно 50%, или между приблизительно 25% и приблизительно 50%,
vi) экспрессия одного или нескольких генов, вовлеченных в фотосинтез, увеличена в растении или его части по сравнению с соответствующим растением дикого типа или его частью,
vii) растение или его часть имеет увеличенное содержание углерода в части или по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью, по меньшей мере приблизительно на 10%, по меньшей мере приблизительно на 25%, по меньшей мере приблизительно на 50%, по меньшей мере приблизительно на 75%, по меньшей мере приблизительно на 100%, по меньшей мере приблизительно на 125%, по меньшей мере приблизительно на 150%, между приблизительно 10% и приблизительно 300%, между приблизительно 50% и приблизительно 250%, или между приблизительно 100% и приблизительно 200%,
viii) растение или его часть имеет увеличенное содержание энергии в части или по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью, по меньшей мере приблизительно на 10%, по меньшей мере приблизительно на 25%, по меньшей мере приблизительно на 50%, по меньшей мере приблизительно на 75%, по меньшей мере приблизительно на 100%, по меньшей мере приблизительно на 125%, по меньшей мере приблизительно на 150%, по меньшей мере приблизительно на 200%, по меньшей мере приблизительно на 250%, между приблизительно 10% и приблизительно 400%, между приблизительно 50% и приблизительно 300%, или между приблизительно 200% и приблизительно 300%,
ix) растение или его часть имеет уменьшенное содержание крахмала в части или по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью, по меньшей мере приблизительно в 2 раза, по меньшей мере приблизительно в 5 раз, по меньшей мере приблизительно в 10 раз, по меньшей мере приблизительно в 15 раз, по меньшей мере приблизительно в 20 раз, по меньшей мере приблизительно в 25 раз, между приблизительно 5-кратным и приблизительно 35-кратным, между приблизительно 10-кратным и приблизительно 30-кратным или между приблизительно 20-кратным и приблизительно 30-кратным,
x) растение или его часть имеет уменьшенное содержание TDF в части или по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью, по меньшей мере приблизительно на 10%, по меньшей мере приблизительно 30%, по меньшей мере приблизительно на 50%, между приблизительно 10% и приблизительно 70%, или между приблизительно 30% и приблизительно 65%, и
xi) растение или его часть имеет содержание растворимого сахара в части или по меньшей мере, в части трансгенного растения, по сравнению с соответствующим растением дикого типа или его частью, составляющее приблизительно от 0,5-кратного до 2-кратного.
В другом варианте осуществления, растение или его часть дополнительно содержат одно или несколько, или все из;
a) первой генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в катаболизм триацилглицеринов (TAG) в растении или его части, предпочтительно, TAG-липазы SDP1, по сравнению с соответствующим растением или его частью, лишенными генетической модификации,
b) третьего экзогенного полинуклеотида, кодирующего полипептид, который увеличивает экспорт жирных кислот из пластид растения, по сравнению с соответствующим растением, лишенным третьего экзогенного полинуклеотида, предпочтительно, ацил-ACP-тиоэстеразу,
c) четвертого экзогенного полинуклеотида, кодирующего второй полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в растении или его части, предпочтительно, полипептид LEC2,
d) пятого экзогенного полинуклеотида, кодирующего полипептид покрытия масляных телец (OBC),
e) второй генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в импорт жирных кислот в пластиды растения, по сравнению с соответствующим растением, лишенным второй генетической модификации, и
f) третьей генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в продукцию диацилглицерина (DAG) в пластиде, по сравнению с соответствующим растением, лишенным третьей генетической модификации, где каждый экзогенный полинуклеотид является функционально связанным с промотором, способным управлять экспрессией полинуклеотида в растении или его части.
В предпочтительном варианте осуществления, присутствие первой генетической модификации, третьего экзогенного полинуклеотида или четвертого экзогенного полинуклеотида, вместе с первым и вторым экзогенными полинуклеотидами увеличивает общее содержание неполярных липидов в растении или его части, предпочтительно, в вегетативной части растения, такой как лист или стебель, по сравнению с соответствующим растением или его частью, которые содержат первый и второй экзогенные полинуклеотиды, но лишены каждого из первой генетической модификации, третьего экзогенного полинуклеотида и четвертого экзогенного полинуклеотида. Более предпочтительно, увеличение является синергическим. Наиболее предпочтительно, по меньшей мере промотор, управляющий экспрессией первого экзогенного полинуклеотида, представляет собой промотор, отличный от конститутивного промотора. Альтернативно, для Sorghum или Zea mays, промотор, предпочтительно, представляет собой конститутивный промотор, например, такой как промотор гена убиквитина.
В одном варианте осуществления, введение одного или нескольких экзогенных полинуклеотидов или генетических модификаций, предпочтительно, экзогенного полинуклеотида, кодирующего OBC или тиоэстеразу жирного ацила, или генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в катаболизм триацилглицеринов (TAG) в растении или его части, более предпочтительно, экзогенного полинуклеотида, кодирующего тиоэстеразу FATA или LDAP, или уменьшающего экспрессию эндогенной TAG-липазы, такой как TAG-липаза SDP1, в растении или его части, приводит к синергическому увеличению общего содержания неполярных липидов в растении или его части, при добавлении к паре трансгенов WRI1 и DGAT, особенно до цветения растения и, даже более конкретно, в стеблях и/или корнях растения. Например, см. Примеры 8, 11 и 15. В предпочтительном варианте осуществления, увеличение содержания TAG в стебле или корне растения, по меньшей мере в 2 раза, более предпочтительно, по меньшей мере в 3 раза больше, чем в соответствующих растении или его части, трансформированных генами, кодирующими WRI1 и DGAT1, но лишенных тиоэстеразы FATA, LDAP и генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в катаболизм триацилглицеринов (TAG) в клетке. Наиболее предпочтительно, по меньшей мере промотор, управляющий экспрессией первого экзогенного полинуклеотида, представляет собой промотор, отличный от конститутивного промотора. Альтернативно, для Sorghum или Zea mays, промотор, предпочтительно, представляет собой конститутивный промотор например, такой как промотор гена убиквитина.
В одном варианте осуществления, каждая генетическая модификация независимо представляет собой мутацию эндогенного гена, которая частично или полностью инактивирует ген, такую как точечная мутация, инсерция или делеция, или экзогенный полинуклеотид, кодирующий молекулу РНК, ингибирующую экспрессию эндогенного гена, где экзогенный полинуклеотид является функционально связанным с промотором, способным управлять экспрессией полинуклеотида в растении или его части. Точечная мутация может представлять собой преждевременный стоп-кодон, мутацию участка сплайсинга, мутацию сдвига рамки или мутацию замены аминокислоты, которая уменьшает активность гена или кодируемого полипептида. Делеция может состоять из одного или нескольких нуклеотидов в пределах транскрибированного экзона или промотора гена, или простираться через экзон или в более чем один экзон, или расширяться до делеции всего гена. Предпочтительно, делецию вводят с использованием технологий ZF, TALEN или CRISPR. В альтернативном варианте осуществления, одна или несколько, или все генетические модификации представляют собой экзогенный полинуклеотид, кодирующий молекулу РНК, уменьшающую экспрессию эндогенного гена, где экзогенный полинуклеотид является функционально связанным с промотором, способным управлять экспрессией полинуклеотида в растении или его части. Примеры экзогенных полинуклеотидов, уменьшающих экспрессию эндогенного гена, выбраны из группы, состоящей из антисмыслового полинуклеотида, смыслового полинуклеотида, микроРНК, полинуклеотида, кодирующего полипептид, который связывается с эндогенным ферментом, молекулой двухцепочечной РНК и полученной из нее процессированной молекулой РНК. В одном варианте осуществления, растение или его часть содержит генетические модификации, которые представляют собой введенную мутацию в эндогенном гене и экзогенный полинуклеотид, кодирующий молекулу РНК, уменьшающего экспрессию другого эндогенного гена. Альтернативно, все из генетических модификаций, обеспечивающих увеличенные TTQ и/или уровни TAG, представляют собой мутации эндогенных генов.
В одном варианте осуществления, растение или его часть имеет одно или несколько, или все из;
i) полипептид фактора транскрипции выбран из группы, состоящей из Wrinkled 1 (WRI1), листовой семядоли 1 (LEC1), LEC1-подобного, листовой семядоли 2 (LEC2), BABY BOOM (BBM), FUS3, ABI3, ABI4, ABI5, Dof4 и Dof11, или из группы, состоящей из MYB73, bZIP53, AGL15, MYB115, MYB118, TANMEI, WUS, GFR2a1, GFR2a2 и PHR1,
ii) полипептид, вовлеченный в биосинтез одного или нескольких неполярных липидов, представляет собой ацилтрансферазу жирных ацилов, вовлеченную в биосинтез TAG, DAG или моноацилглицерина (MAG) в растении или его части, такой как DGAT, PDAT, LPAAT, GPAT или MGAT, предпочтительно, полипептида DGAT или PDAT, или PDCT или CPT, или полипептида PLC или PLD,
iii) полипептид, вовлеченный в катаболизм триацилглицеринов (TAG) в растении или его части, представляет собой липазу SDP1, полипептид Cgi58, ацил-CoA-оксидазу, такую как ACX1 или ACX2, или полипептид, вовлеченный в β-окисление жирных кислот в растении или его части, такой как пероксисомальный транспортер АТФ-связывающей кассеты PXA1, предпочтительно, липаза SDP1,
iv) полипептид покрытия масляных телец (OBC) представляет собой олеозин, такой как полиолеозин или калеозин, или ассоциированный с липидными капельками белок (LDAP), предпочтительно, неаллергенный OBC,
v) полипептид, который увеличивает экспорт жирных кислот из пластид в растении или его части, представляет собой тиоэстеразу C16 или C18 жирных кислот, такую как полипептид FATA или полипептид FATB, транспортер жирных кислот, такой как полипептид ABCA9, или длинноцепочечную ацил-CoA-синтетазу (LACS),
vi) полипептид, вовлеченный в импорт жирных кислот в пластиды в растении или его части, представляет собой транспортер жирных кислот или его субъединицу, предпочтительно, полипептид TGD, и
vii) полипептид, вовлеченный в продукцию диацилглицерина (DAG) в пластиде, представляет собой пластидную GPAT, пластидную LPAAT или пластидную PAP.
В одном варианте осуществления, активность PDCT или CPT в клетке или вегетативной части растения увеличена, по сравнению с клеткой или вегетативной частью растения дикого типа. Альтернативно, активность PDCT или CPT уменьшена, например, посредством мутации в эндогенном гене, кодирующем фермент, или посредством понижающей регуляции гена посредством молекулы РНК, которая уменьшает его экспрессию.
В одном варианте осуществления, полипептид, вовлеченный в биосинтез одного или нескольких неполярных липидов, представляет собой DGAT или PDAT, и полипептид, вовлеченный в катаболизм TAG в растении или его части представляет собой липазу SDP1.
В одном варианте осуществления, полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в растении или его части, представляет собой полипептид WRI1, и полипептид, вовлеченный в биосинтез одного или нескольких неполярных липидов, представляет собой DGAT или PDAT.
В одном варианте осуществления, полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в растении или его части, представляет собой полипептид WRI1, полипептид LEC2, полипептид LEC1 или LEC1-подобный полипептид, и полипептид, вовлеченный в биосинтез одного или нескольких неполярных липидов, представляет собой DGAT.
В одном варианте осуществления, полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в растении или его части, представляет собой полипептид WRI1, полипептид LEC2, полипептид LEC1 или LEC1-подобный полипептид, и полипептид, вовлеченный в катаболизм триацилглицеринов (TAG) в растении или его части, представляет собой липазу SDP1.
В одном варианте осуществления, полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в растении или его части, представляет собой полипептид WRI1, полипептид LEC2, полипептид LEC1 или LEC1-подобный полипептид, полипептид, вовлеченный в биосинтез одного или нескольких неполярных липидов, представляет собой DGAT или PDAT, и полипептид, вовлеченный в катаболизм триацилглицеринов (TAG) в растении или его части, представляет собой липазу SDP1.
В одном варианте осуществления, полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в растении, представляет собой полипептид WRI1, полипептид LEC2, полипептид LEC1 или LEC1-подобный полипептид, и полипептид, вовлеченный в импорт жирных кислот в пластиды в растении или его части, представляет собой полипептид TGD.
В одном варианте осуществления, полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в растении, представляет собой полипептид WRI1, полипептид LEC2, полипептид LEC1 или LEC1-подобный полипептид, и полипептид, вовлеченный в продукцию диацилглицерина (DAG), представляет собой пластидную GPAT.
В одном варианте осуществления, полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в растении, представляет собой полипептид WRI1, полипептид LEC2, полипептид LEC1 или LEC1-подобный полипептид, полипептид, который увеличивает экспорт жирных кислот из пластид растения представляет собой тиоэстеразу жирных кислот, предпочтительно, полипептид FATA или FATB, и полипептид, вовлеченный в импорт жирных кислот в пластиды растения, представляет собой полипептид TGD.
В одном варианте осуществления, полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в растении, представляет собой полипептид WRI1, полипептид LEC2, полипептид LEC1 или LEC1-подобный полипептид, полипептид, который увеличивает экспорт жирных кислот из пластид растения представляет собой тиоэстеразу жирных кислот, предпочтительно, полипептид FATA или FATB, и полипептид, вовлеченный в продукцию диацилглицерина (DAG), представляет собой пластидную GPAT.
В одном варианте осуществления, полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в растении, представляет собой полипептид WRI1, полипептид LEC2, полипептид LEC1 или LEC1-подобный полипептид, полипептид, вовлеченный в импорт жирных кислот в пластиды растения, представляет собой полипептид TGD, и полипептид, вовлеченный в продукцию диацилглицерина (DAG), представляет собой пластидную GPAT.
В одном варианте осуществления, когда они присутствуют, два фактора транскрипции представляют собой WRI1 и LEC2, или WRI1 и LEC1.
В вышеуказанных вариантах осуществления, растение или его часть предпочтительно, содержит экзогенный полинуклеотид, кодирующий DGAT, и генетическую модификацию, осуществляющую понижающую регуляцию продукции эндогенной липазы SDP1. Более предпочтительно, растение или его часть не содержит экзогенного полинуклеотида, кодирующего PDAT, и/или представляет собой растение или его часть, отличные от Nicotiana benthamiana или его части, и/или WRI1 представляет собой WRI1, отличный от WRI1 Arabidopsis thaliana (SEQ ID NO:21 или 22), и/или представляет собой растение или его часть, отличные от Brassica napus или его части. В одном варианте осуществления, по меньшей мере один из экзогенных полинуклеотидов в растении или его части экспрессируется с промотора, который не является конститутивным промотором например, такого как промотор, который экспрессируется предпочтительно, в зеленых тканях или стеблях растения, или который подвергается повышающей регуляции после начала цветения или в ходе старения.
В одном варианте осуществления, экзогенный полинуклеотид, кодирующий WRI1, содержит одно или несколько из следующего:
i) нуклеотидов, кодирующих полипептид, содержащий аминокислоты, последовательность которых указана как любая из SEQ ID NO:21-75 или 196-201, или его биологически активный фрагмент, или полипептид, аминокислотная последовательность которого является по меньшей мере на 30% идентичной любой одной или нескольким из SEQ ID NO: 21-75 или 196-201,
ii) нуклеотидов, последовательность которых является по меньшей мере на 30% идентичной i), и
iii) нуклеотидов, которые гибридизуются с i) и/или ii) в строгих условиях. Предпочтительно, полипептид WRI1 представляет собой полипептид WRI1, отличный от WRI1 Arabidopsis thaliana (SEQ ID NO:21 или 22). Более предпочтительно, полипептид WRI1 содержит аминокислоты, последовательность которых указана как SEQ ID NO:199, или его биологически активный фрагмент, или полипептид, аминокислотная последовательность которого является по меньшей мере на 30% идентичной этому.
В одном варианте осуществления, часть представляет собой вегетативную часть, и один или несколько, или все из промоторов экспрессируются на более высоком уровне в вегетативной части, по сравнению с семенем растения.
В следующем варианте осуществления, растение или его часть имеет одно или несколько, или все из;
i) растение или его часть имеет общее содержание неполярных липидов по меньшей мере приблизительно 8%, по меньшей мере приблизительно 10%, по меньшей мере приблизительно 11%, по меньшей мере приблизительно 12%, по меньшей мере приблизительно 15%, по меньшей мере приблизительно 20%, по меньшей мере приблизительно 25%, по меньшей мере приблизительно 30%, по меньшей мере приблизительно 35%, по меньшей мере приблизительно 40%, по меньшей мере приблизительно 45%, по меньшей мере приблизительно 50%, по меньшей мере приблизительно 55%, по меньшей мере приблизительно 60%, по меньшей мере приблизительно 65%, по меньшей мере приблизительно 70%, между 8% и 75%, между 10% и 75%, между 11% и 75%, между приблизительно 15% и 75%, между приблизительно 20% и 75%, между приблизительно 30% и 75%, между приблизительно 40% и 75%, между приблизительно 50% и 75%, между приблизительно 60% и 75%, или между приблизительно 25% и 50% (масс./масс. сухой массы), предпочтительно, до цветения,
ii) вегетативная часть растения имеет содержание TAG по меньшей мере приблизительно 8%, по меньшей мере приблизительно на 10%, по меньшей мере приблизительно 11%, по меньшей мере приблизительно 12%, по меньшей мере приблизительно 15%, по меньшей мере приблизительно 20%, по меньшей мере приблизительно 25%, по меньшей мере приблизительно 30%, по меньшей мере приблизительно 35%, по меньшей мере приблизительно 40%, по меньшей мере приблизительно 45%, по меньшей мере приблизительно 50%, по меньшей мере приблизительно 55%, по меньшей мере приблизительно 60%, по меньшей мере приблизительно 65%, по меньшей мере приблизительно 70%, между 8% и 75%, между 10% и 75%, между 11% и 75%, между приблизительно 15% и 75%, между приблизительно 20% и 75%, между приблизительно 30% и 75%, между приблизительно 40% и 75%, между приблизительно 50% и 75%, между приблизительно 60% и 75%, или между приблизительно 25% и 50% (масс./масс. сухой массы), предпочтительно, до цветения,
iii) один или несколько, или все из промоторов выбраны из тканеспецифического промотора, такого как специфический для листьев и/или стеблей промотор, регулируемого развитием промотора, такого как специфический для старения промотор, например, промотор SAG12, индуцибельного промотора, или регулируемого суточным ритмом промотора,
iv) растение или его часть является одним членом популяции или коллекции из по меньшей мере приблизительно 1500, из по меньшей мере приблизительно 3000 или из по меньшей мере приблизительно 5000 таких растений или их частей, предпочтительно, вегетативных частей растений, где первый и второй экзогенные полинуклеотиды вставлены в одной и той же хромосомной локализации в геноме каждого из растений,
v) растение является членом семейства Fabaceae (или Leguminosae), например, люцерна, клевер, горох, люцерна посевная, фасоль, чечевица, люпин, мескито, кэроб, соя и арахис, или членом семейства Poaceae, например, кукуруза или сорго, и
vi) часть представляет собой лист или листья, которые являются зрелыми.
В одном варианте осуществления, до цветения растения, вегетативная часть растения имеет общее содержание неполярных липидов по меньшей мере приблизительно 8%, по меньшей мере приблизительно 10%, приблизительно 11%, между 8% и 15%, или между 9% и 12% (масс./масс. сухой массы).
В следующем варианте осуществления, растение или его часть представляет собой;
i) растение 16:3 или его вегетативную часть, или семя, и при этом содержит одно или несколько, или все из следующего:
a) экзогенного полинуклеотида, кодирующего полипептид, который увеличивает экспорт жирных кислот из пластид растения, по сравнению с соответствующим растением, лишенным экзогенного полинуклеотида,
b) первой генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в импорт жирных кислот в пластиды растения, по сравнению с соответствующим растением, лишенным первой генетической модификации, и
c) второй генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в продукцию диацилглицерина (DAG) в пластиде, по сравнению с соответствующим растением, лишенным второй генетической модификации,
где экзогенный полинуклеотид является функционально связанным с промотором, способным управлять экспрессией полинуклеотида в растении или его части, или
ii) растение 18:3 или его вегетативную часть, или семя.
В одном варианте осуществления, растение или его часть имеет одно или несколько, или все из;
i) растение содержит часть, предпочтительно, вегетативную часть, имеющую увеличенный синтез общих жирных кислот, по сравнению с соответствующей частью, лишенной первого экзогенного полинуклеотида, или уменьшенный катаболизм общих жирных кислот, по сравнению с соответствующей частью, лишенной первого экзогенного полинуклеотида, или и то, и другое, так что она имеет увеличенный уровень общих жирных кислот, по сравнению с соответствующей частью, лишенной первого экзогенного полинуклеотида,
ii) растение содержит часть, предпочтительно, вегетативную часть, имеющую увеличенную экспрессию и/или активность ацилтрансферазы жирных ацилов, катализирующей синтез TAG, DAG или MAG, предпочтительно, TAG, по сравнению с соответствующей частью, имеющей первый экзогенный полинуклеотид и лишенной экзогенного полинуклеотида, который кодирует полипептид, вовлеченный в биосинтез одного или нескольких неполярных липидов,
iii) растение содержит часть, предпочтительно, вегетативную часть, имеющую уменьшенную продукцию лизофосфатидиновой кислоты (LPA) из ацил-ACP и G3P в пластидах, по сравнению с соответствующей частью, имеющей первый экзогенный полинуклеотид и лишенной генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в продукцию диацилглицерина (DAG) в пластидах в части растения,
iv) растение содержит часть, предпочтительно, вегетативную часть, имеющую измененное соотношение C16:3 и C18:3 жирных кислот в общем содержании жирных кислот и/или содержание галактолипидов, по сравнению с соответствующей частью, лишенной экзогенного полинуклеотида(полинуклеотидов) и/или генетической модификации(модификаций), предпочтительно, уменьшенное соотношение,
v) олеиновая кислота составляет по меньшей мере 20% (моль %), по меньшей мере 22% (моль %), по меньшей мере 30% (моль %), по меньшей мере 40% (моль %), по меньшей мере 50% (моль %), или по меньшей мере 60% (моль %), предпочтительно, приблизительно 65% (моль %), или между 20% и приблизительно 65% от общего содержания жирных кислот в растении или его части,
vi) неполярный липид в растении или его части предпочтительно, вегетативной части, имеет увеличенный уровень одной или нескольких жирных кислот, которые содержат гидроксильную группу, эпоксигруппу, циклопропановую группу, двойную углерод-углеродную связь, тройную углерод-углеродную связь, конъюгированные двойные связи, разветвленную цепь, такую как метилированная или гидроксилированная разветвленная цепь, или комбинацию двух или более из них, или любую из двух, трех, четырех, пяти или шести вышеупомянутых групп, связей или разветвленных цепей,
vii) неполярный липид в растении или его части предпочтительно, вегетативной части, содержит одну или несколько полиненасыщенных жирных кислот, выбранных из эйкозадиеновой кислоты (EDA), арахидоновой кислоты (ARA), стеаридоновой кислоты (SDA), эйкозатриеновой кислоты (ETE), эйкозатетраеновой кислоты (ETA), эйкозапентаеновой кислоты (EPA), докозапентаеновой кислоты (DPA), докозагексаеновой кислоты (DHA), или комбинации двух или более из них,
viii) часть представляет собой вегетативную часть растения, такую как лист или стебель, или их часть,
ix) один или несколько, или все из промоторов выбраны из промотора, отличного от конститутивного промотора, предпочтительно, тканеспецифического промотора, такого как специфический для листьев и/или стеблей промотор, регулируемого развитием промотора, такого как специфический для старения промотор, например, промотор SAG12, индуцибельного промотора, или регулируемого суточным ритмом промотора, предпочтительно, где по меньшей мере один из промоторов, функционально связанных с экзогенным полинуклеотидом, кодирующим полипептид фактора транскрипции, представляет собой промотор, отличный от конститутивного промотора,
x) растение или его часть, предпочтительно, вегетативная часть, имеют общее содержание жирных кислот, уровень олеиновой кислоты и/или уровень пальмитиновой кислоты в котором увеличен по меньшей мере на 2%, по сравнению с соответствующим растением или его частью, лишенными экзогенного полинуклеотида(полинуклеотидов) и/или генетической модификации(модификаций), и/или уровень α-линоленовой кислоты (ALA) и/или уровень линолеиновой кислоты в котором уменьшен по меньшей мере на 2%, по сравнению с соответствующим растением или его частью, лишенными экзогенного полинуклеотида(полинуклеотидов) и/или генетической модификации(модификаций),
xi) неполярный липид в растении или его части предпочтительно, вегетативной части, включает модифицированный уровень общих стеролов, предпочтительно, свободных (неэтерифицированных) стеролов, стероильных сложных эфиров, стероилгликозидов, по сравнению с неполярным липидом в соответствующем растении или его части, лишенных экзогенного полинуклеотида(полинуклеотидов) и/или генетической модификации(модификаций),
xii) неполярный липид в растении или его части содержит воски и/или сложные эфиры восков,
xiii) растение содержит экзогенный полинуклеотид, кодирующий супрессор сайленсинга, где экзогенный полинуклеотид является функционально связанным с промотором, способным управлять экспрессией полинуклеотида в растении,
xiv) уровень одного или нескольких неполярных липида(липидов) и/или общее содержание неполярных липидов в растении или его части, предпочтительно, вегетативной части растения, по меньшей мере на 2% масс. больше, чем в соответствующем растении или части, соответственно, содержащих экзогенные полинуклеотиды, кодирующие WRI1 Arabidposis thaliana (SEQ ID NO:21) и DGAT1 Arabidopsis thaliana (SEQ ID NO:1),
xv) общее содержание полиненасыщенных жирных кислот (PUFA), уменьшенное относительно общего содержания PUFA в соответствующем растении, лишенном экзогенного полинуклеотида(полинуклеотидов) и/или генетической модификации(модификаций),
xvi) часть растения представляет собой клубень картофеля (Solanum tuberosum), клубень сахарной свеклы (Beta vulgaris), стебель сахарного тростника (вида Saccharum) или сорго (Sorghum bicolor), семя однодольного растения, имеющее увеличенное общее содержание жирных кислот в эндосперме например, такое как зерно пшеницы (Triticum aestivum) или семя кукурузы (Zea mays), лист видов Nicotiana или семя бобовых, имеющее увеличенное общее содержание жирных кислот например, такое как семя вида Brassica или семя сои (Glycine max),
xvii) если часть растения представляет собой семя, семя прорастает со скоростью, по существу такой же, как для соответствующего семени дикого типа, или после посева в грунт образует растение, семя которого прорастает со скоростью, по существу такой же, как для соответствующего семени дикого типа, и
xviii) растение представляет собой растение водорослей, например, диатомовых (бациллариофитов), зеленых водорослей (хлорофитов), сине-зеленых водорослей (цианофитов), золотисто-коричневых водорослей (хризофитов), гаптофитов, коричневых водорослей или водорослей-гетероконтов.
В одном варианте осуществления, растение или его часть содержит первый экзогенный полинуклеотид, кодирующий WRI1, второй экзогенный полинуклеотид, кодирующий DGAT или PDAT, предпочтительно, DGAT1, третий экзогенный полинуклеотид, кодирующий РНК, которая уменьшает экспрессию гена, кодирующего полипептид SDP1, и четвертый экзогенный полинуклеотид, кодирующий олеозин. В предпочтительных вариантах осуществления, растение или его часть имеет один или несколько, или все из следующих признаков:
i) общее содержание липидов по меньшей мере 8%, по меньшей мере 10%, по меньшей мере 12%, по меньшей мере 14% или по меньшей мере 15,5% (% сухой массы),
ii) по меньшей мере в 3 раза, по меньшей мере в 5 раз, по меньшей мере в 7 раз, по меньшей мере в 8 раз или по меньшей мере в 10 раз более высокое общее содержание липидов в растении или его части, по сравнению с соответствующим растением или его частью, лишенными экзогенных полинуклеотидов и генетических модификаций,
iii) общее содержание TAG по меньшей мере 5%, по меньшей мере 6%, по меньшей мере 6,5% или по меньшей мере 7% (% масс. сухой массы или массы семени),
iv) по меньшей мере в 40 раз, по меньшей мере в 50 раз, по меньшей мере в 60 раз или по меньшей мере в 70 раз, по меньшей мере в 100 раз, или по меньшей мере в 120 раз более высокое общее содержание TAG, по сравнению с соответствующим растением или его частью, лишенных экзогенных полинуклеотидов и генетических модификаций,
v) олеиновая кислота составляет по меньшей мере 15%, по меньшей мере 19% или по меньшей мере 22% (% масс. сухой массы или массы семени) жирных кислот в TAG,
vi) по меньшей мере в 10 раз, по меньшей мере в 15 раз или по меньшей мере в 17 раз более высокий уровень олеиновой кислоты в TAG, по сравнению с соответствующим растением или его частью, лишенных экзогенных полинуклеотидов и генетических модификаций,
vii) пальмитиновая кислота составляет по меньшей мере 20%, по меньшей мере 25%, по меньшей мере 30% или по меньшей мере 33% (% масс.) жирных кислот в TAG,
viii) по меньшей мере в 1,5 раз более высокий уровень пальмитиновой кислоты in TAG, по сравнению с соответствующим растением или его частью, лишенных экзогенных полинуклеотидов и генетических модификаций,
ix) линолеиновая кислота составляет по меньшей мере 22%, по меньшей мере 25%, по меньшей мере 30% или по меньшей мере 34% (% масс.) жирных кислот в TAG,
x) α-линоленовая кислота составляет менее чем 20%, менее чем 15%, менее чем 11% или менее чем 8% (% масс.) жирных кислот в TAG,
xi) по меньшей мере в 5 раз, или по меньшей мере в 8 раз более низкий уровень α-линоленовой кислоты в TAG по сравнению с соответствующим растением или его частью, лишенных экзогенных полинуклеотидов и генетических модификаций, и
xii) когда часть представляет собой клубень картофеля, содержание TAG составляет по меньшей мере 0,5% в пересчете на сухую массу, и/или общее содержание жирных кислот составляет по меньшей мере 1%, предпочтительно, по меньшей мере 1,5% или по меньшей мере 2,0%, в пересчете на сухую массу.
В вышеуказанных вариантах осуществления, предпочтительной частью растения является фрагмент листа с площадью поверхности по меньшей мере 1 см2 или фрагмент стебля длиной по меньшей мере 1 см.
В одном варианте осуществления вышеуказанных аспектов, растение или часть растения обрабатывают таким образом, что они становятся неспособными размножаться или дать начало живому растению, т.е. они являются мертвыми, например, бурые листья или стебель. Например, растение или часть растения высушивают и/или перемалывают. В другом варианте осуществления, часть растения является живой, например, зеленый лист или стебель.
В одном варианте осуществления, часть представляет собой семя, плод или вегетативную часть, такую как воздушная часть растения или зеленая часть, такая как лист или стебель.
В вышеуказанных вариантах осуществления, является предпочтительным, чтобы часть представляла собой вегетативную часть растения, которое выращивают в грунте, или которое выращено в грунте, и затем часть растения собирают, и где вегетативная часть содержит по меньшей мере 8% TAG в пересчете на массу (% сухой массы), например, между 8% и 75% или между 8% и 30%. Более предпочтительно, содержание TAG составляет по меньшей мере 10%, например, между 10% и 75% или между 10% и 30%. Предпочтительно, эти уровни TAG присутствуют в вегетативных частях до или во время цветения растения, или до стадии завязывания семян. В этих вариантах осуществления, является предпочтительным, чтобы соотношение содержание TAG в листьях к содержанию TAG в стеблях растения составляет между 1:1 и 10:1, и/или соотношение увеличено по сравнению с соответствующей вегетативной частью, содержащей первый и второй экзогенные полинуклеотиды и лишенной первой генетической модификации. Предпочтительно, вегетативная часть растения имеет увеличенное содержание растворимого белка, по сравнению с соответствующей вегетативной частью дикого типа, по меньшей мере приблизительно на 100% или между приблизительно 50% и приблизительно 125%. Предпочтительно, вегетативная часть растения имеет увеличенное содержание азота, по сравнению с соответствующей вегетативной частью дикого типа, по меньшей мере приблизительно на 100%, или между приблизительно 50% и приблизительно 125%. Предпочтительно, вегетативная часть растения имеет уменьшенное соотношение углерод:азот, по сравнению с соответствующей вегетативной частью растения дикого типа, по меньшей мере приблизительно на 40%, или между приблизительно 25% и приблизительно 50%. Предпочтительно, вегетативная часть растения имеет уменьшенное содержание TDF, по сравнению с соответствующей вегетативной частью растения дикого типа, по меньшей мере приблизительно на 30% или между приблизительно 30% и приблизительно 65%.
В одном варианте осуществления, растение по изобретению представляет собой однодольное растение или его часть, предпочтительно, лист, семя, стебель, корень или эндосперм, имеющим общее содержание жирных кислот или содержание TAG, увеличенное по меньшей мере в 5 раз в пересчете на массу, по сравнению с соответствующим однодольным растением дикого типа или его частью. Альтернативно, однодольное растение содержит эндосперм, имеющий содержание TAG, составляющее по меньшей мере 2,0%, предпочтительно, по меньшей мере 3%, более предпочтительно, по меньшей мере 4% или по меньшей мере 5%, в пересчете на массу части растения, предпочтительно, листа, стебля, корня, зерна или эндосперма. В одном варианте осуществления, эндосперм имеет содержание TAG по меньшей мере 2%, что по меньшей мере в 5 раз увеличено по сравнению с соответствующим эндоспермом дикого типа. Предпочтительно, растение имеет полную мужскую и женскую фертильность, его пыльца является по существу на 100% жизнеспособной, и его семя имеет скорость прорастания, составляющую между 70% и 100%, по сравнению с соответствующим семенем дикого типа. В одном варианте осуществления, трансгенное растение по изобретению представляет собой растение-потомок, по меньшей мере через два поколения от начального трансгенного растения, и предпочтительно, является гомозиготным по трансгенам. В вариантах осуществления, однодольное растение или его часть, предпочтительно, лист, стебель, семя или эндосперм, дополнительно отличается одним или несколькими признаками, как описано в контексте растения или его части по изобретению. В вариантах осуществления, однодольное растение или его часть, предпочтительно, лист, зерно, стебель или эндосперм, по изобретению, предпочтительно, имеет увеличенный уровень мононенасыщенных жирных кислот (MUFA) и/или уменьшенный уровень полиненасыщенных жирных кислот (PUFA) как в общем содержании жирных кислот, так и в фракции TAG от общего содержания жирных кислот, например, такой как увеличенный уровень олеиновой кислоты и уменьшенный уровень LA (18:2), по сравнению с соответствующим растением или его частью, лишенными генетических модификаций и/или экзогенного полинуклеотида(полинуклеотидов). Предпочтительно, уровень линолеиновой кислоты (LA, 18:2) в общем содержании жирных кислот в семени или эндосперме однодольного растения уменьшен по меньшей мере на 5%, и/или уровень олеиновой кислоты в общем содержании жирных кислот увеличен по меньшей мере на 5% по сравнению с соответствующим растением дикого типа или его частью, предпочтительно, по меньшей мере 10% или более предпочтительно, по меньшей мере 15%, по сравнению с соответствующим растением или его частью, лишенными генетических модификаций и/или экзогенного полинуклеотида(полинуклеотидов).
В одном варианте осуществления, растение или его часть представляет собой Acrocomia aculeata (макаубская пальма), Arabidopsis thaliana, Aracinis hypogaea (арахис), Astrocaryum murumuru (мурумуру), Astrocaryum vulgare (тукума), Attalea geraensis (атталия), Attalea humilis (американская масличная пальма), Attalea oleifera (андайя), Attalea phalerata (урикури), Attalea speciosa (бабассу), Avena sativa (овес), Beta vulgaris (сахарная свекла), виды Brassica, например, Brassica carinata, Brassica juncea, Brassica napobrassica, Brassica napus (рапс), Camelina sativa (ложный лен), Cannabis sativa (конопля), Carthamus tinctorius (сафлор красильный), Caryocar brasiliense (пекви), Cocos nucifera (кокосовый орех), Crambe abyssinica (абиссинская капуста), Cucumis melo (дыня), Elaeis guineensis (африканская пальма), Glycine max (соя), Gossypium hirsutum (хлопчатник), виды Helianthu, например, Helianthus annuus (подсолнечник), Hordeum vulgare (ячмень), Jatropha curcas (лечебный орех), Joannesia princeps (лещина арара), виды Lemna (ряска), такие как Lemna aequinoctialis, Lemna disperma, Lemna ecuadoriensis, Lemna gibba (ряска горбатая), Lemna japonica, Lemna minor, Lemna minuta, Lemna obscura, Lemna paucicostata, Lemna perpusilla, Lemna tenera, Lemna trisulca, Lemna turionifera, Lemna valdiviana, Lemna yungensis, Licania rigida (ойтика), Linum usitatissimum (лен), Lupinus angustifolius (люпин), Mauritia flexuosa (пальма бурити), Maximiliana maripa (пальма инайя), виды Miscanthus, такие как Miscanthus x giganteus и Miscanthus sinensis, виды Nicotiana (табак), такие как Nicotiana tabacum или Nicotiana benthamiana, Oenocarpus bacaba (бакаба-до-азейте), Oenocarpus bataua (патайя), Oenocarpus distichus (бакаба-де-лек), виды Oryza (рис), такие как Oryza sativa и Oryza glaberrima, Panicum virgatum (просо прутьевидное), Paraqueiba paraensis (марь), Persea amencana (авокадо), Pongamia pinnata (индийский бук), Populus trichocarpa, Ricinus communis (клещевина обыкновенная), виды Saccharum (сахарный тростник), Sesamum indicum (кунжут), Solanum tuberosum (картофель), виды Sorghum, например, Sorghum bicolor, Sorghum vulgare, Theobroma grandiforum (какао), виды Trifolium, Trithrinax brasiliensis (бразильская игольчатая пальма), виды Triticum (пшеница),Triticum aestivum и Zea mays (кукуруза).
В одном варианте осуществления, растение или его часть является одним членом популяции или коллекции из по меньшей мере приблизительно 1500, из по меньшей мере приблизительно 3000 или из по меньшей мере приблизительно 5000 таких растений или их частей.
В одном варианте осуществления, содержание TFA, содержание TAG, общее содержание неполярных липидов или одного или нескольких неполярных липидов, и/или уровень олеиновой кислоты или PUFA в растении или его части поддается определению посредством анализа с использованием газовой хроматографии метиловых сложных эфиров жирных кислот, полученных из растения или его вегетативной части.
В следующем варианте осуществления, часть растения представляет собой лист, и общее содержание неполярных липидов в листе поддается определению посредством анализа с использованием ядерного магнитного резонанса (ЯМР).
В каждом из вышеуказанных вариантов осуществления, является предпочтительным, чтобы растение представляло собой трансгенное растение-потомок, по меньшей мере через два поколения от начального трансгенного растения, и предпочтительно, является гомозиготным по трансгенам.
В одном варианте осуществления, растение или его часть по изобретению является фенотипически нормальным, в том смысле, что его способность к росту и воспроизводству значительно не уменьшена, по сравнению с немодифицированным растением или его частью. В одном варианте осуществления, биомасса, скорость роста, скорость прорастания, размер запасающего органа, размер семени и/или количество образовавшихся жизнеспособных семян составляет не менее 70%, не менее 80% или не менее 90% от показателей соответствующего растения дикого типа, при выращивании в идентичных условиях. В одном варианте осуществления, растение имеет мужскую и женскую фертильность в той же степени, что и соответствующее растение дикого типа, а его пыльца (если она образуется) так же жизнеспособна, как и пыльца соответствующего растения дикого типа, предпочтительно, является жизнеспособной по меньшей мере приблизительно на 75% или по меньшей мере приблизительно на 90%, или близко к 100%. В одном варианте осуществления, растение образует семя, имеющее скорость прорастания по меньшей мере приблизительно 75% или по меньшей мере приблизительно 90%, относительно скорости прорастания соответствующего семени растения дикого типа, если вид растений образует семена. В одном варианте осуществления, растение по изобретению имеет высоту растения, составляющую по меньшей мере приблизительно 75% или по меньшей мере приблизительно 80%, или по меньшей мере приблизительно 90% относительно высоты соответствующего растения дикого типа, выращенного в таких же условиях. Предусмотрена комбинация каждого из указанных признаков. В альтернативном варианте осуществления, высота растения по изобретению составляет между 60% и 90% относительно высоты соответствующего растения дикого типа, выращенного в таких же условиях. В одном варианте осуществления, растение или его часть по изобретению, предпочтительно, лист растения, не проявляет усиленного некроза, т.е. степень некроза, если он присутствует, такая же, какую имеет соответствующее растение дикого типа или его часть, выращенное в таких же условиях и находящееся на такой же стадии развития растения. Этот признак применим, в частности, к растению или его части, содержащему экзогенный полинуклеотид, кодирующий тиоэстеразу жирных кислот, такую как тиоэстераза FATB.
В следующем аспекте, настоящее изобретение относится к коллекции из по меньшей мере приблизительно 1500, по меньшей мере приблизительно 3000 или по меньшей мере приблизительно 5000 вегетативных частей растений, где каждая является вегетативной частью растения по изобретению, где вегетативные части растений собраны из растений, растущих в поле.
В одном варианте осуществления, первый и второй экзогенные полинуклеотиды вставлены в одной и той же хромосомной локализации в геноме каждой из вегетативных частей растений, предпочтительно, в ядерном геноме каждой из вегетативных частей растений.
Настоящее изобретение относится также к закрому для хранения, содержащему коллекцию вегетативных частей растений по изобретению.
Настоящее изобретение далее относится к семени из, или полученному из, растения по изобретению, предпочтительно, к коллекции из по меньшей мере приблизительно 1500, по меньшей мере приблизительно 3000, по меньшей мере приблизительно 5000, или по меньшей мере приблизительно 10000 семян по изобретению, содержащих экзогенные полинуклеотиды.
В другом аспекте настоящее изобретение относится к экстракту растения или его части по изобретению. Экстракт, предпочтительно, имеет другой состав жирных кислот, по сравнению с соответствующим экстрактом дикого типа.
В одном варианте осуществления, экстракт содержит первый и второй экзогенные полинуклеотиды.
В одном варианте осуществления, экстракт лишен по меньшей мере 50% или по меньшей мере 90% неполярных липидов из растения или его части.
В одном варианте осуществления, экстракт включает содержание растворимого белка из растения или его части.
В одном варианте осуществления, экстракт включает содержание азота из растения или его части.
В одном варианте осуществления, экстракт лишен по меньшей мере 50% или по меньшей мере 90% хлорофилла и/или растворимых сахаров из растения или его части.
В одном варианте осуществления, экстракт включает содержание углерода из растения или его части.
В одном варианте осуществления, экстракт содержит краситель, связывающий белок в экстракте.
Экстракты по изобретению можно легко получать с использованием стандартных в данной области способов.
Комбинации признаков из вышеуказанных аспектов явно предусмотрены для растения, части растения и клетки по изобретению, и в способах их получения и применения.
В другом аспекте настоящее изобретение относится к способу получения экстракта растения, включающему
i) получение растения или его части по изобретению, или семени по изобретению, и
ii) обработку растения или его части, или семени, для получения экстракта.
В одном варианте осуществления, растение или его часть по изобретению, или семя по изобретению, является трансгенным.
В одном варианте осуществления, стадия ii) включает получение двух или более фракций из растения или его части, или семени, и отбор по меньшей мере одной, но не всех, из фракций.
В одном варианте осуществления, отобранная фракция(фракции) имеет один или несколько из следующих признаков;
i) содержит первый и второй экзогенные полинуклеотиды,
ii) лишена по меньшей мере 50% или по меньшей мере 90% неполярных липидов из растения или его части,
iii) включает содержание растворимого белка из растения или его части,
iv) включает содержание азота из растения или его части,
v) лишена по меньшей мере 50% или по меньшей мере 90% хлорофилла и/или растворимых сахаров из растения или его части, и
vi) включает содержание углерода из растения или его части.
В следующем аспекте, настоящее изобретение относится к способу отбора растения или его части с желательным фенотипом, включающему
i) получение множества растений-кандидатов или их частей, где каждое содержит
a) первый экзогенный полинуклеотид, кодирующий полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в растении или его части, и
b) второй экзогенный полинуклеотид, кодирующий полипептид, вовлеченный в биосинтез одного или нескольких неполярных липидов,
где каждый экзогенный полинуклеотид является функционально связанным с промотором, способным управлять экспрессией полинуклеотида в растении или его части,
ii) анализ липидов во множестве частей, или по меньшей мере, в части каждого растения во множестве растений-кандидатов, со стадии i),
iii) анализ во множестве частей, или по меньшей мере, в части каждого растения во множестве растений-кандидатов, со стадии i) одного или нескольких, или всех из;
a) содержания растворимого белка,
b) содержания азота,
c) соотношения углерод:азот,
d) экспрессии фотосинтетического гена,
e) способности к фотосинтезу,
f) общего содержания пищевых волокон (TDF),
g) содержания углерода,
h) содержания энергии,
i) содержания TAG, и
j) TTQ, и
iv) отбор растения или его части, имеющего увеличенное TTQ и/или увеличенное содержание триацилглицерина (TAG) в части или по меньшей мере части растения по сравнению с соответствующим растением дикого типа или его частью, и желательный фенотип, выбранный из одного или нескольких, или всего из следующего;
A) увеличенного содержания растворимого белка в части или по меньшей мере части растения по сравнению с соответствующим растением дикого типа или его частью,
B) увеличенного содержания азота в части или по меньшей мере части растения по сравнению с соответствующим растением дикого типа или его частью,
C) уменьшенного соотношения углерод:азот в части или по меньшей мере части растения по сравнению с соответствующим растением дикого типа или его частью,
D) увеличенной экспрессии фотосинтетического гена в части или по меньшей мере части растения по сравнению с соответствующим растением дикого типа или его частью,
E) увеличенной способности к фотосинтезу в части или по меньшей мере части растения по сравнению с соответствующим растением дикого типа или его частью,
F) уменьшенного общего содержания пищевых волокон (TDF) в части или по меньшей мере части растения по сравнению с соответствующим растением дикого типа или его частью,
G) увеличенного содержание углерода в части или по меньшей мере части растения по сравнению с соответствующим растением дикого типа или его частью, и
H) увеличенного содержания энергии в части или по меньшей мере части растения по сравнению с соответствующим растением дикого типа или его частью.
В одном варианте осуществления, увеличенное содержание триацилглицерина (TAG) определяют посредством анализа одного или нескольких из общего содержания жирных кислот, содержания TAG, состава жирных кислот, любыми способами, которые могут включать или могут не включать сначала экстракцию липида.
В другом варианте осуществления, отобранное растение или его часть имеет один или несколько признаков, как описано в настоящем описании.
В другом аспекте настоящее изобретение относится к способу получения пищевого продукта, включающего смешивание растения или его части из любого растения по изобретению, семени по изобретению или экстракта по изобретению, по меньшей мере с одним другим пищевым ингредиентом.
В другом аспекте настоящее изобретение относится к пищевому продукту, содержащему клетку по изобретению, растение или его часть по изобретению, семя по изобретению, экстрагированное масло или экстракт по изобретению.
В одном варианте осуществления, пищевой продукт представляет собой силос, гранулы или сено.
В другом аспекте, настоящее изобретение относится к способу кормления животного, включающему предоставление животному клетки по изобретению, растения или его части по изобретению, семени по изобретению, экстрагированного липида или другого экстракта по изобретению, или пищевого продукта по изобретению. В одном варианте осуществления, материал, предоставленный животному, представляет собой остаточный растительный материал, остающийся после того, как по меньшей мере некоторое количество масла было экстрагировано, такой как жмыховая мука или листовая/стеблевая мука.
В одном варианте осуществления, животное употребляет внутрь увеличенное количество азота, белка, углерода и/или энергетического потенциала относительно того, когда животное употребляет внутрь такое же количество, в пересчете на сухую массу, соответствующей клетки дикого типа, растения или его части, семени или экстракта, или пищевого продукта, полученного из соответствующего растения или его части дикого типа.
Авторы настоящего изобретения идентифицировали подкласс OBC, которые являются неаллергенными, или неизвестными как аллергенными, например, для человека.
Таким образом, в следующем аспекте, настоящее изобретение относится к рекомбинантной эукариотической клетке, предпочтительно, клетке вида Sorghum или Zea mays, содержащей по меньшей мере первый экзогенный полинуклеотид, кодирующий неаллергенный OBC, где экзогенный полинуклеотид является функционально связанным с промотором, способным управлять экспрессией полинуклеотида в клетке.
В одном варианте осуществления, первый экзогенный полинуклеотид содержит одно или несколько из следующего:
i) нуклеотидов, кодирующих полипептид, содержащий аминокислоты, последовательность которых указана как любая из SEQ ID NO: 306-314, или его биологически активный фрагмент, или полипептид OBC, аминокислотная последовательность которого является по меньшей мере на 30% идентичной любой одной или нескольким из SEQ ID NO: 306-314, где полипептид OBC является неаллергенным,
ii) нуклеотидов, последовательность которых является по меньшей мере на 30% идентичной i), и
iii) полинуклеотида, который гибридизуется с одним или обоими из i) или ii) в строгих условиях. В одном варианте осуществления, олеозин L не представляет собой олеозин L кунжута (SEQ ID NO:305).
В одном варианте осуществления, рекомбинантная клетка содержит одно или несколько из следующего:
a) второго экзогенного полинуклеотида, кодирующего полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в клетке, предпочтительно, полипептид WRI1,
b) третьего экзогенного полинуклеотида, кодирующего полипептид, вовлеченный в биосинтез одного или нескольких неполярных липидов, предпочтительно, DGAT и/или PDAT,
c) первой генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в катаболизм триацилглицеринов (TAG) в клетке, предпочтительно, TAG-липазы SDP1, по сравнению с соответствующей клеткой, лишенной генетической модификации,
d) четвертого экзогенного полинуклеотида, кодирующего полипептид, который увеличивает экспорт жирных кислот из пластид клетки, по сравнению с соответствующей клеткой, лишенной экзогенного полинуклеотида, предпочтительно, полипептид ацил-ACP-тиоэстеразы,
e) пятого экзогенного полинуклеотида, кодирующего второй полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в клетке, предпочтительно, полипептид LEC2,
f) второй генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в импорт жирных кислот в пластиды клетки, предпочтительно, полипептида TGD, по сравнению с соответствующей клеткой, лишенной генетической модификации,
g) шестого экзогенного полинуклеотида, кодирующего ассоциированный с липидными капельками белок (LDAP),
h) третьей генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в продукцию диацилглицерина (DAG) в пластиде, по сравнению с соответствующей клеткой, лишенной генетической модификации, и
i) четвертой генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в импорт жирных кислот в пластиды клетки, по сравнению с соответствующей клеткой, лишенной генетической модификации,
где каждый экзогенный полинуклеотид является функционально связанным с промотором, способным управлять экспрессией полинуклеотида в клетке.
В одном варианте осуществления, клетка представляет собой клетку растения, полученную из или находящуюся в вегетативной части растения, и один или несколько, или все из промоторов экспрессируются на более высоком уровне в вегетативной части, по сравнению с семенем растения.
В одном варианте осуществления, первый экзогенный полинуклеотид является кодон-оптимизированным для экспрессии в клетке растения, такой как клетка вида Sorghum или Zea mays.
В одном варианте осуществления, используют один или несколько, или все из следующих признаков вышеуказанных аспектов:
i) клетка имеет увеличенный синтез общих жирных кислот, по сравнению с соответствующей клеткой, лишенной второго экзогенного полинуклеотида, или уменьшенный катаболизм общих жирных кислот, по сравнению с соответствующей клеткой, лишенной второго экзогенного полинуклеотида, или оба, таким образом, что она имеет увеличенный уровень общих жирных кислот, по сравнению с соответствующей клеткой, лишенной второго экзогенного полинуклеотида,
ii) клетка имеет увеличенную экспрессию и/или активность ацилтрансферазы жирных ацилов, катализирующей синтез TAG, DAG или MAG, предпочтительно, TAG, по сравнению с соответствующей клеткой, содержащей второй экзогенный полинуклеотид и лишенной третьего экзогенного полинуклеотида, кодирующего полипептид, вовлеченный в биосинтез одного или нескольких неполярных липидов,
iii) клетка имеет уменьшенную продукцию лизофосфатидиновой кислоты (LPA) from ацил-ACP и G3P в пластидах, по сравнению с соответствующей клеткой, содержащей второй экзогенный полинуклеотид и лишенной третьей генетической модификации, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в продукцию диацилглицерина (DAG) в пластиде, в клетке,
iv) клетка имеет измененное соотношение C16:3 и C18:3 жирных кислот в общем содержании жирных кислот и/или содержании галактолипидов, по сравнению с соответствующей клеткой, лишенной экзогенного полинуклеотида(полинуклеотидов) и/или генетической модификации(модификаций), предпочтительно, уменьшенное соотношение,
v) клетка находится в вегетативной части растения и имеет общее содержание неполярных липидов по меньшей мере приблизительно 8%, по меньшей мере приблизительно 10%, по меньшей мере приблизительно 11%, по меньшей мере приблизительно 12%, по меньшей мере приблизительно 15%, по меньшей мере приблизительно 20%, по меньшей мере приблизительно 25%, по меньшей мере приблизительно 30%, по меньшей мере приблизительно 35%, по меньшей мере приблизительно 40%, по меньшей мере приблизительно 45%, по меньшей мере приблизительно 50%, по меньшей мере приблизительно 55%, по меньшей мере приблизительно 60%, по меньшей мере приблизительно 65%, по меньшей мере приблизительно 70%, между 8% и 75%, между 10% и 75%, между 11% и 75%, между приблизительно 15% и 75%, между приблизительно 20% и 75%, между приблизительно 30% и 75%, между приблизительно 40% и 75%, между приблизительно 50% и 75%, между приблизительно 60% и 75%, или между приблизительно 25% и 50% (масс./масс. сухой массы),
vi) клетка находится в вегетативной части растения и имеет содержание TAG по меньшей мере приблизительно 8%, по меньшей мере приблизительно 10%, по меньшей мере приблизительно 11%, по меньшей мере приблизительно 12%, по меньшей мере приблизительно 15%, по меньшей мере приблизительно 20%, по меньшей мере приблизительно 25%, по меньшей мере приблизительно 30%, по меньшей мере приблизительно 35%, по меньшей мере приблизительно 40%, по меньшей мере приблизительно 45%, по меньшей мере приблизительно 50%, по меньшей мере приблизительно 55%, по меньшей мере приблизительно 60%, по меньшей мере приблизительно 65%, по меньшей мере приблизительно 70%, между 8% и 75%, между 10% и 75%, между 11% и 75%, между приблизительно 15% и 75%, между приблизительно 20% и 75%, между приблизительно 30% и 75%, между приблизительно 40% и 75%, между приблизительно 50% и 75%, между приблизительно 60% и 75%, или между приблизительно 25% и 50% (масс./масс. сухой массы),
vii) полипептид фактора транскрипции выбран из группы, состоящей из Wrinkled 1 (WRI1), листовой семядоли 1 (LEC1), LEC1-подобного, листовой семядоли 2 (LEC2), BABY BOOM (BBM), FUS3, ABI3, ABI4, ABI5, Dof4 и Dof11,
viii) олеиновая кислота составляет по меньшей мере 20% (моль %), по меньшей мере 22% (моль %), по меньшей мере 30% (моль %), по меньшей мере 40% (моль %), по меньшей мере 50% (моль %), или по меньшей мере 60% (моль %), предпочтительно, приблизительно 65% (моль %) или между 20% и приблизительно 65% от общего содержания жирных кислот в клетке,
ix) неполярный липид в клетке содержит жирную кислоту, которая содержит гидроксильную группу, эпоксигруппу, циклопропановую группу, двойную углерод-углеродную связь, тройную углерод-углеродную связь, конъюгированные двойные связи, разветвленную цепь, такую как метилированная или гидроксилированная разветвленная цепь, или комбинацию двух или более из них, или любую из двух, трех, четырех, пяти или шести вышеупомянутых групп, связей или разветвленных цепей,
x) неполярный липид в клетке содержит одну или несколько полиненасыщенных жирных кислот, выбранных из эйкозадиеновой кислоты (EDA), арахидоновой кислоты (ARA), стеаридоновой кислоты (SDA), эйкозатриеновой кислоты (ETE), эйкозатетраеновой кислоты (ETA), эйкозапентаеновой кислоты (EPA), докозапентаеновой кислоты (DPA), докозагексаеновой кислоты (DHA) или комбинации двух или более из них,
xi) клетка находится в растении или его части, предпочтительно, вегетативной части растения, или клетка представляет собой клетку водорослей, например, диатомовых (бациллариофитов), зеленых водорослей (хлорофитов), сине-зеленых водорослей (цианофитов), золотисто-коричневых водорослей (хризофитов), гаптофитов, коричневых водорослей или водорослей-гетероконтов, или клетка получена из микроорганизма, или представляет собой микроорганизм, подходящий для ферментации, такой как гриб,
xii) один или несколько, или все из промоторов выбраны из тканеспецифического промотора, такого как специфический для листьев и/или стеблей промотор, регулируемого развитием промотора, такого как специфический для старения промотор, например, промотор SAG12, индуцибельного промотора или регулируемого суточным ритмом промотора,
xiii) клетка имеет общее содержание жирных кислот, включающее среднецепочечные жирные кислоты, предпочтительно, C12:0, C14:0 или обе, на уровне по меньшей мере 5% от общего содержания жирных кислот, и необязательно экзогенный полинуклеотид, кодирующий LPAAT, которая проявляет преимущественную активность в отношении жирных кислот со сресуток длиной цепи (C8-C14), предпочтительно, C12:0 или C14:0,
xiv) клетка имеет общее содержание жирных кислот, уровень олеиновой кислоты в котором увеличен по меньшей мере на 2%, по сравнению с соответствующей клеткой, лишенной экзогенного полинуклеотида(полинуклеотидов) и/или генетической модификации(модификаций), и/или уровень α-линоленовой кислоты (ALA) в котором уменьшен по меньшей мере на 2%, по сравнению с соответствующей клеткой, лишенной экзогенного полинуклеотида(полинуклеотидов) и/или генетической модификации(модификаций),
xv) неполярный липид в клетке включает модифицированный уровень общих стеролов, предпочтительно, свободных (неэтерифицированных) стеролов, стероильных сложных эфиров, стероилгликозидов, по сравнению с неполярным липидом в соответствующей клетке, лишенной экзогенного полинуклеотида(полинуклеотидов) и/или генетической модификации(модификаций),
xvi) неполярный липид в клетке содержит воски и/или сложные эфиры восков,
xvii) клетка является одним из членов популяции или коллекции из по меньшей мере приблизительно 1000 таких клеток, предпочтительно, в вегетативной части растения или семени,
xviii) клетка содержит экзогенный полинуклеотид, кодирующий супрессор сайленсинга, где экзогенный полинуклеотид является функционально связанным с промотором, способным управлять экспрессией полинуклеотида в клетке,
xix) уровень одного или нескольких неполярных липида(липидов) и/или общее содержание неполярных липидов в клетке по меньшей мере на 2% масс. больше, чем в соответствующей клетке, содержащей экзогенные полинуклеотиды, кодирующие WRI1 Arabidposis thaliana (SEQ ID NO:21) и DGAT1 Arabidopsis thaliana (SEQ ID NO:1), и
xx) общее содержание полиненасыщенных жирных кислот (PUFA), уменьшенное относительно общего содержания PUFA в соответствующей клетке, лишенной экзогенного полинуклеотида(полинуклеотидов) и/или генетической модификации(модификаций).
Любой вариант осуществления в настоящем описании следует принимать как применимый, с необходимыми изменениями, к любому другому варианту осуществления, если конкретно не указано иное.
Комбинации признаков из вышеуказанных аспектов явно предусмотрены для растения, части растения и клетки по изобретению, и в способах их получения и применения.
Объем настоящего изобретения не является ограниченным конкретными вариантами осуществления, описанными в настоящем описании, которые предназначены только для целей иллюстрации. Функционально эквивалентные продукты, композиции и способы явно включены в объем изобретения, как описано в настоящем описании.
На протяжении этого описания, если конкретно не указано иное или контекст не требует иного, ссылку на одну стадию, состав вещества, группу стадий или группу составов вещества следует принимать как включающую единственное и множественное число (т.е. одно или несколько) из этих стадий, составов вещества, групп стадий или групп составов вещества.
Изобретение далее описано посредством следующих неограничивающих примеров и со ссылкой на прилагаемые фигуры.
КРАТКОЕ ОПИСАНИЕ СОПУТСТВУЮЩИХ ЧЕРТЕЖЕЙ
Фигура 1. Схема синтеза липида в эукариотических клетках, демонстрирующая экспорт некоторых из жирных кислот, синтезированных в пластидах, в эндоплазматический ретикулум (ER) через связанную с пластидой мембрану (PLAM), и импорт некоторых из жирных кислот из ER в пластиду для эукариотического синтеза галактолипидов. Сокращения:
ацетил-CoA и малонил-CoA: ацетил-кофермент A и малонил-кофермент A;
ACCаза: ацетил-CoA карбоксилаза;
FAS: комплекс синтазы жирных кислот;
16:0-ACP, 18:0-ACP и 18:1-ACP: C16:0-ацилпереносящий белок (ACP), C18:0-ацилпереносящий белок, C18:1-ацилпереносящий белок;
KAS II: кетоацил-ACP синтетаза II (EC 2.3.1.41);
PLPAAT: пластидная LPAAT;
PGPAT: пластидная GPAT;
PAP: фосфорилаза PA (EC 3.1.3.4);
G3P: глицерин-3-фосфат;
LPA: лизофосфатидиновая кислота;
PA: фосфатидиновая кислота;
DAG: диацилглицерин;
TAG: триацилглицерин;
ацил-CoA и ацил-PC: ацил-кофермент A и ацил-фосфатидилхолин;
PC: фосфатидилхолин;
GPAT: глицерил-3-фосфат-ацилтрансфераза;
LPAAT: ацилтрансфераза лизофосфатидиновой кислоты (EC 2.3.1.51);
LPCAT: ацил-CoA:лизофосфатидилхолин-ацилтрансфераза; или синонимы 1-ацилглицерофосфохолин-O-ацилтрансфераза; ацил-CoA:1-ацил-sn-глицеро-3-фосфохолин O-ацилтрансфераза (EC 2.3.1.23),
CPT: CDP-холин:диацилглицерил-холинфосфотрансфераза; или синонимы 1-алкил-2-ацетилглицерил-холинфосфотрансфераза; алкилацилглицерил-холинфосфотрансфераза; холинфосфотрансфераза; фосфорилхолин-глицерид трансфераза (EC 2.7.8.2),
PDCT: фосфатидилхолин:диацилглицерил-холинфосфотрансфераза,
PLC: фосфолипаза C (EC 3.1.4.3),
PLD: фосфолипаза D; холинфосфатаза; лецитиназа D; липофосфодиэстераза II (EC 3.1.4.4),
PDAT: фосфолипид: диацилглисерил-ацилтрансфераза; или синоним фосфолипид: 1,2-диацил-sn-глицерил-O-ацилтрансфераза (EC 2.3.1.158);
FAD2: Δ12-десатураза жирных кислот; FAD3, Δ15-десатураза жирных кислот;
УДФ-гал: уридиндифосфатгалактоза;
MGDS: моногалактозилдиацилглицерин-синтетаза;
MGDG: моногалактозилдиацилглицерин; DGDG: дигалактозилдиацилглицерин
FAD6, 7, 8: пластидная Δ12-десатураза жирных кислот, пластидная ω3-десатураза, пластидная ω3-десатураза, индуцируемые при низкой температуре, соответственно.
Фигура 2. Схематическое представление конструкции шпильки N. benthamiana SDP1. Проиллюстрированные генетические фрагменты являются такими, как описано в Примере 2. Сокращения являются такими, как для Фигуры 12. Сайты attB представляют собой сайты рекомбинации из вектора pHELLSGATE12.
Фигура 3. Содержание TAG в образцах зеленых листьев растений табака, трансформированных T-ДНК из pOIL51, линии №61 и №69, собранных до цветения. Контрольные (родительские) образцы были получены от растений, трансформированных T-ДНК из pJP3502.
Фигура 4. Уровни TAG (% сухой массы) в ткани корня и стебля растений дикого типа (wt) и трансгенных растений N. tabacum, содержащих только T-ДНК из pJP3502 или дополнительно содержащих T-ДНК из pOIL051.
Фигура 5. Содержание TAG в образцах листьев трансформированных растений табака в стадии роста с завязыванием семян, трансформированных T-ДНК из pOIL049, линии №23c и №32b. Контрольные (родительские) образцы были получены от растений, трансформированных T-ДНК из pJP3502. Верхняя линия иллюстрирует процент 18:2 в TAG, а нижняя линия иллюстрирует процент 18:3 (ALA) в содержании жирных кислот.
Фигура 6. Уровни TAG (% сухой массы) в ткани корня и стебля растений дикого типа (wt) и трансгенных растений N. tabacum содержащих только T-ДНК из pJP3502 или дополнительно содержащих T-ДНК из pOIL049.
Фигура 7. A. Содержание крахмала в ткани листа растений дикого типа (WT) и трансгенных растений, содержащих T-ДНК из pJP3502 (контроль HO) или T-ДНК как из pJP3502, так и из pOIL051 (pOIL51.61 и pOIL51.69), или как из pJP3502, так и из pOIL049 (pOIL49.32b). Данные представляют объединенные результаты по меньшей мере для трех индивидуальных растений. B. Корреляция между содержанием крахмала и TAG в ткани листа растений дикого типа (WT) и трансгенных растений, содержащих T-ДНК из pJP3502 (контроль HO) или T-ДНК как из pJP3502, так и из pOIL051 (pOIL51.61 и pOIL51.69), или как из pJP3502, так и из pOIL049 (pOIL49.32b). Данные представляют объединенные результаты по меньшей мере для трех индивидуальных растений.
Фигура 8. Содержание крахмала и растворимого сахара в пересчете на сухую массу (DW) в стареющих листьях растений дикого типа (пустые круги) и трансгенных растений (закрашенные круги) (T1), образцы которых отобраны на стадии завязывания семян. Трансгенные растения N. tabacum включают растения, обозначенные HO, SDP1 и LEC2. В каждом случает три растения включены в анализ. Точки данных основаны на анализах в трех повторах.
Фигура 9. Содержание N (A) и растворимого белка (B) в листьях WT и HO различного возраста, собранных с растений на 69 DAS.
Фигура 10. Содержание растворимого белка в листьях табака WT и HO как функция возраста листьев и растений.
Фигура 11. Среднее общее содержание жирных кислот (TFA) в мг/100 мг сухой массы листьев 9, 15 и 20 в растениях табака, выращенных в модифицированных условиях: увеличенная интенсивность освещения (верхняя левая панель); контроль (верхняя правая панель); увеличенный фотопериод, увеличенная интенсивность освещения и увеличенная концентрация CO2 (нижняя левая панель); уменьшенный фотопериод при высокой интенсивности освещения (нижняя правая панель).
Фигура 12. Разделение TLC общих липидов листьев, экстрагированных из S. Bicolor дикого типа и трансгенного S. bicolor. Wt, дикий тип; EV, контроль с пустым вектором; 2, S. bicolor, трансформированный pOIL136 (событие 2); TAG, триацилглицерин; FFA, свободные жирные кислоты; DAG, диацилглицерин. Ткани листа собирали с молодых, вегетативных растений после переноса в грунт.
Фигура 13. A. Уровни липидов в листьях сорго, трансформированных с использованием комбинации генетических конструкций pOIL103 и pOIL197, на вегетативной стадии роста. Показаны уровни (% масс. сухой массы) TFA, TAG и полярных липидов. Каждая группа из 4 столбцов показывает, по порядку, уровни в листьях из растений дикого типа (WT, синий), контрольных растений с пустым вектором (EV, оранжевый) и трансгенных растений TX-03-8 (серый) и TX-03-38 (желтый). B. Уровни галактолипидов MGDG и DGDG и фосфолипидов PG, PC, PE, PA, PS и PI в листьях, как для A.
Фигура 14. Схематическая диаграмма вектора pOIL122. Сокращения: TER Agrtu-NOS, терминатор нопалинсинтетазы Agrobacterium tumefaciens; NPTII, кодирующая область белка неомицинфосфотрансферазы; PRO CaMV35S-Ex2, промотор 35S вируса мозаики цветной капусты с двойным участком энхансера; Arath-DGAT1, кодирующая область белка ацилтрансферазы DGAT1 Arabidopsis thaliana; PRO Arath-Rubisco SSU, промотор малой субъединицы Rubisco A. thaliana; Arath-FATA2, кодирующая область белка тиоэстеразы FATA2 A. thaliana; Arath-WRI, кодирующая область белка фактора транскрипции WRI1 A. thaliana; TER Glyma-Lectin, терминатор лектина Glycine max; промотор enTCUP2, криптический конститутивный промотор Nicotiana tabacum; attB1 и attB2, сайты рекомбинации Gateway; фрагмент NB SDP1, участок SDP1 Nicotiana benthamiana, служащий мишенью для сайленсинга шпРНКи; терминатор ОКС, терминатор октопинсинтетазы A. tumefaciens. Признаки остова за пределами участка T-ДНК получены из pORE04 (Coutu et al., 2007).
Фигура 15. Уровни TAG (% сухой массы листа) в ткани листа N. benthamiana, инфильтрованной генами, кодирующими различные полипептиды WRI1, либо в присутствии (столбцы справа), либо в отсутствие (столбцы слева) соэкспрессии DGAT1 (n=3). Все образцы были инфильтрованы также конструкцией P19.
Фигура 16. Филогенетическое дерево полипептидов LDAP (Пример 11).
Фигура 17. Схематическое представление генетической конструкции pJP3506, включая участок T-ДНК между левой и правой границами. Сокращения такие же, как для Фигуры 12, и: Sesin-олеозин, кодирующая область белка олеозина Sesame indicum.
Фигура 18. Содержание жирных кислот трансгенных семян пшеницы.
Фигура 19. Уровни TFA и TAG (% масс. сухой массы листа) в листьях растений сорго на стадии роста трубчатого листа, для растений дикого типа (отрицательный контроль), растений, трансформированных генетической конструкцией для экспрессии DGAT и олеозина (DGAT+олеозин), растений, трансформированных генетической конструкцией для экспрессии WRI, экспрессированного с промотора Ubi (Ubi::WRI1), растений, трансформированных генетическими конструкциями для экспрессии DGAT, олеозина и WRI, экспрессированных с промотора Ubi (Ubi::WRI1+DGAT+олеозин), растений, трансформированных генетическими конструкциями для экспрессии DGAT, олеозина и WRI, экспрессированных с промотора PEPC (PEPC::WRI1+DGAT+олеозин), растений, трансформированных генетическими конструкциями для экспрессии DGAT, олеозина и WRI, экспрессированных с промотора SSU (SSU::WRI1+DGAT+олеозин). Каждая точка представляет уровни, наблюдаемые для независимого трансгенного растения. Для каждого типа растений, столбцы точек слева (синий) показывают уровни TFA, и столбцы точек (красный) показывают уровни TAG в такой же группе растений.
Фигура 20. Содержание TAG и состав жирных кислот для избранных жирных кислот в тканях листьев N. benthamiana после введения генов, кодирующих WRI1, DGAT1 и полипептид масляных телец (pOIL382-387).
КЛЮЧ К СПИСКУ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
SEQ ID NO: 1 полипептид DGAT1 Arabidopsis thaliana (CAB44774.1)
SEQ ID NO: 2 полипептид DGAT2 Arabidopsis thaliana (NP_566952.1)
SEQ ID NO: 3 полипептид DGAT2 Ricinus communis (AAY16324.1)
SEQ ID NO: 4 полипептид DGAT2 Vernicia fordii (ABC94474.1)
SEQ ID NO: 5 полипептид DGAT2 Mortierella ramanniana (AAK84179.1)
SEQ ID NO: 6 полипептид DGAT2 Homo sapiens (Q96PD7.2)
SEQ ID NO: 7 полипептид DGAT2 Homo sapiens (Q58HT5.1)
SEQ ID NO: 8 полипептид DGAT2 Bos taurus (Q70VZ8.1)
SEQ ID NO: 9 полипептид DGAT2 Mus musculus (AAK84175.1)
SEQ ID NO: 10 YFP трипептид-консервативная DGAT2 и/или мотив последовательности MGAT1/2
SEQ ID NO: 11 HPHG тетрапептид-консервативная DGAT2 и/или мотив последовательности MGAT1/2
SEQ ID NO: 12 мотив последовательности EPHS тетрапептид-консервативной растительной DGAT2
SEQ ID NO: 13 RXGFX(K/R)XAXXXGXXX(L/V)VPXXXFG(E/Q) -длинный консервативный мотив последовательности DGAT2, который представляет собой часть предполагаемого глицерил-фосфолипидного домена
SEQ ID NO: 14 FLXLXXXN - консервативный мотив последовательности DGAT2 и MGAT1/2 мыши, который представляет собой предполагаемый домен связывания с нейтральным липидом
SEQ ID NO: 15 домен plsC ацилтрансферазы (PF01553) GPAT
SEQ ID NO: 16 домен суперсемейства галогенокислоты дегалогеназы (HAD)-подобной гидролазы (PF12710) GPAT
SEQ ID NO: 17 домен фосфосерин фосфатазы (PF00702). GPAT4-8 содержит N-концевой участок, гомологичный указанному домену.
SEQ ID NO: 18 Консервативная последовательность аминокислот GPAT GDLVICPEGTTCREP
SEQ ID NO: 19 Консервативная последовательность аминокислот GPAT/фосфатазы (Мотив I)
SEQ ID NO: 20 Консервативная последовательность аминокислот GPAT/фосфатазы (Мотив III)
SEQ ID NO: 21 полипептид WRI1 Arabidopsis thaliana (A8MS57).
SEQ ID NO: 22 полипептид WRI1 Arabidopsis thaliana (Q6X5Y6)
SEQ ID NO: 23 полипептид WRI1 Arabidopsis lyrata подвид lyrata (XP_002876251.1)
SEQ ID NO: 24 полипептид WRI1 Brassica napus (ABD16282.1)
SEQ ID NO: 25полипептид WRI1Brassica napus (ADO16346.1)
SEQ ID NO: 26 полипептид WRI1 Glycine max (XP_003530370.1)
]SEQ ID NO: 27 полипептид WRI1 Jatropha curcas (AEO22131.1)
[SEQ ID NO: 28 полипептид WRI1 Ricinus communis (XP_002525305.1)
SEQ ID NO: 29 полипептид WRI1 Populus trichocarpa (XP_002316459.1)
SEQ ID NO:30 полипептид WRI1 Vitis vinifera (CBI29147.3)
SEQ ID NO: 31 полипептид WRI1 Brachypodium distachyon (XP_003578997.1)
SEQ ID NO: 32 полипептид WRI1 Hordeum vulgare подвид. vulgare (BAJ86627.1)
SEQ ID NO: 33 полипептид WRI1 Oryza sativa (EAY79792.1)
SEQ ID NO: 34 полипептид WRI1 Sorghum bicolor (XP_002450194.1)
SEQ ID NO: 35 полипептид WRI1 Zea mays (ACG32367.1)
SEQ ID NO: 36 полипептид WRI1 Brachypodium distachyon (XP_003561189.1)
SEQ ID NO: 37 полипептид WRI1 Brachypodium sylvaticum (ABL85061.1)
SEQ ID NO: 38 полипептид WRI1 Oryza sativa (BAD68417.1)
SEQ ID NO: 39 полипептид WRI1 Sorghum bicolor (XP_002437819.1)
SEQ ID NO: 40 полипептид WRI1 Sorghum bicolor (XP_002441444.1)
SEQ ID NO: 41 полипептид WRI1 Glycine max (XP_003530686.1)
SEQ ID NO: 42 полипептид WRI1 Glycine max (XP_003553203,1)
SEQ ID NO: 43 полипептид WRI1 Populus trichocarpa (XP_002315794.1)
SEQ ID NO: 44 полипептид WRI1 Vitis vinifera (XP_002270149.1)
SEQ ID NO: 45 полипептид WRI1 Glycine max (XP_003533548.1)
SEQ ID NO: 46 полипептид WRI1 Glycine max (XP_003551723.1)
SEQ ID NO: 47 полипептид WRI1 Medicago truncatula (XP_003621117.1)
SEQ ID NO: 48 полипептид WRI1 Populus trichocarpa (XP_002323836.1)
SEQ ID NO: 49 полипептид WRI1 Ricinus communis (XP_002517474.1)
SEQ ID NO: 50 полипептид WRI1 Vitis vinifera (CAN79925.1)
SEQ ID NO: 51 полипептид WRI1 Brachypodium distachyon (XP_003572236.1)
SEQ ID NO: 52 полипептид WRI1 Oryza sativa (BAD10030.1)
SEQ ID NO: 53 полипептид WRI1 Sorghum bicolor (XP_002444429.1)
SEQ ID NO: 54 полипептид WRI1 Zea mays (NP_001170359.1)
SEQ ID NO: 55 полипептид WRI1 Arabidopsis lyrata подвид lyrata (XP_002889265.1)
SEQ ID NO: 56 полипептид WRI1 Arabidopsis thaliana (AAF68121.1)
SEQ ID NO: 57 полипептид WRI1 Arabidopsis thaliana (NP_178088.2)
SEQ ID NO: 55 полипептид WRI1 Arabidopsis lyrata подвид lyrata (XP_002889265.1)
SEQ ID NO: 59 полипептид WRI1 Thellungiella halophila (BAJ33872.1)
SEQ ID NO: 60 полипептид WRI1 Arabidopsis thaliana (NP_563990.1)
SEQ ID NO: 61 полипептид WRI1Glycine max (XP_003530350.1)
SEQ ID NO: 62 полипептид WRI1 Brachypodium distachyon (XP_003578142.1)
SEQ ID NO: 63 полипептид WRI1 Oryza sativa (EAZ09147.1)
SEQ ID NO: 64 полипептид WRI1 Sorghum bicolor (XP_002460236.1)
SEQ ID NO: 65 полипептид WRI1 Zea mays (NP_001146338.1)
SEQ ID NO: 66 полипептид WRI1 Glycine max (XP_003519167.1)
SEQ ID NO: 67 полипептид WRI1 Glycine max (XP_003550676.1)
SEQ ID NO: 68 полипептид WRI1 Medicago truncatula (XP_003610261.1)
SEQ ID NO: 69 полипептид WRI1 Glycine max (XP_003524030.1)
SEQ ID NO: 70 полипептид WRI1 Glycine max (XP_003525949.1)
SEQ ID NO: 71 полипептид WRI1 Populus trichocarpa (XP_002325111.1)
SEQ ID NO: 72 полипептид WRI1 Vitis vinifera (CBI36586.3)
SEQ ID NO: 73 полипептид WRI1 Vitis vinifera (XP_002273046.2)
SEQ ID NO: 74 полипептид WRI1 Populus trichocarpa (XP_002303866.1)
SEQ ID NO: 75 полипептид WRI1 Vitis vinifera (CBI25261.3)
SEQ ID NO: 76 Sorbi-WRL1
SEQ ID NO: 77 Lupan-WRL1
SEQ ID NO: 78 Ricco-WRL1
SEQ ID NO: 79 полипептид WRI1 Lupin angustifolius
SEQ ID NO: 80 полипептид DGAT1 Aspergillus fumigatus (XP_755172.1)
SEQ ID NO: 81 полипептид DGAT1 Ricinus communis (AAR11479.1)
SEQ ID NO: 82 полипептид DGAT1 Vernicia fordii (ABC94472.1)
SEQ ID NO: 83 полипептид DGAT1 Vernonia galamensis (ABV21945.1)
SEQ ID NO: 84 полипептид DGAT1 Vernonia galamensis (ABV21946.1)
SEQ ID NO: 85 полипептид DGAT1 Euonymus alatus (AAV31083.1)
SEQ ID NO: 86 полипептид DGAT1 Caenorhabditis elegans (AAF82410.1)
SEQ ID NO: 87 полипептид DGAT1 Rattus norvegicus (NP_445889.1)
SEQ ID NO: 88 полипептид DGAT1 Homo sapiens (NP_036211.2)
SEQ ID NO: 89 мотив WRI1 (R G V T/S R H R W T G R)
SEQ ID NO: 90 мотив WRI1 (F/Y E A H L W D K)
SEQ ID NO: 91 мотив WRI1 (D L A A L K Y W G)
SEQ ID NO: 92 мотив WRI1 (S X G F S/A R G X)
SEQ ID NO: 93 мотив WRI1 (H H H/Q N G R/K W E A R I G R/K V)
SEQ ID NO: 94 мотив WRI1 (Q E E A A A X Y D)
SEQ ID NO: 95 полипептид олеозин Brassica napus (CAA57545.1)
SEQ ID NO: 96 полипептид олеозин S1-1Brassica napus (ACG69504.1)
SEQ ID NO: 97 полипептид олеозин S2-1 Brassica napus (ACG69503.1)
SEQ ID NO: 98 полипептид олеозин S3-1 Brassica napus (ACG69513.1)
SEQ ID NO: 99 полипептид олеозин S4-1 Brassica napus (ACG69507.1)
SEQ ID NO: 100 полипептид олеозин S5-1 Brassica napus (ACG69511.1)
SEQ ID NO: 101 полипептид олеозин 1 Arachis hypogaea (AAZ20276.1)
SEQ ID NO: 102 полипептид олеозин 2 Arachis hypogaea (AAU21500.1)
SEQ ID NO: 103 полипептид олеозин 3 Arachis hypogaea (AAU21501.1)
SEQ ID NO: 104 полипептид олеозин 5 Arachis hypogaea (ABC96763.1)
SEQ ID NO: 105 полипептид олеозин 1 Ricinus communis (EEF40948.1)
SEQ ID NO: 106 полипептид олеозин 2 Ricinus communis (EEF51616.1)
SEQ ID NO: 107 полипептид олеозин Glycine max изоформа a (P29530.2)
SEQ ID NO: 108 полипептид олеозин Glycine max изоформа b (P29531.1)
SEQ ID NO: 109 полипептид олеозин Linum usitatissimum низкомолекулярная изоформа (ABB01622.1)
SEQ ID NO: 110 последовательность аминокислот полипептида олеозин Linum usitatissimum высокомолекулярная изоформа (ABB01624.1)
SEQ ID NO: 111 полипептид олеозин Helianthus annuus (CAA44224.1)
SEQ ID NO: 112 полипептид олеозин Zea mays (NP_001105338.1)
SEQ ID NO: 113 полипептид стеролеозин Brassica napus (ABM30178.1)
SEQ ID NO: 114 полипептид стеролеозин SLO1-1 Brassica napus (ACG69522.1)
SEQ ID NO: 115 полипептид стеролеозин SLO2-1 Brassica napus (ACG69525.1)
SEQ ID NO: 116 полипептид стеролеозин Sesamum indicum (AAL13315.1)
SEQ ID NO: 117 полипептид стеролеозин Zea mays (NP_001152614.1)
SEQ ID NO: 118 полипептид калеозин CLO-1 Brassica napus (ACG69529.1)
SEQ ID NO: 119 полипептид калеозин CLO-3 Brassica napus (ACG69527.1)
SEQ ID NO: 120 полипептид калеозин Sesamum indicum (AAF13743.1)
SEQ ID NO: 121 полипептид калеозин Zea mays (NP_001151906.1)
SEQ ID NO: 122 последовательность Т-ДНК pJP3502 (вставленная в геном)
SEQ ID NO: 123 последовательность вектора pJP3507
SEQ ID NO: 124 последовательность линкера
SEQ ID NO: 125 частичная последовательность CGI-58 Nicotiana benthamiana, выбранная для сайленсинга шпРНКи (pTV46)
SEQ ID NO: 126 частичная последовательность АГФазы N. tabacum, выбранная для сайленсинга шпРНКи (pTV35)
SEQ ID NO: 127 мотив липазы GXSXG
SEQ ID NO: 128 мотив ацилтрансферазы HX(4)D
SEQ ID NO: 129 вероятный мотив связывания с липидом VX(3)HGF
SEQ ID NO: 130 полинуклеотид CGi58 Arabidopsis thaliana (NM_118548.1)
SEQ ID NO: 131 полинуклеотид CGi58 Brachypodium distachyon (XM_003578402.1)
SEQ ID NO: 132 полинуклеотид CGi58 Glycine max (XM_003523590.1)
SEQ ID NO: 133 полинуклеотид CGi58 Zea mays (NM_001155541.1)
SEQ ID NO: 134 полинуклеотид CGi58 Sorghum bicolor (XM_002460493.1)
SEQ ID NO: 135 полинуклеотид CGi58 Ricinus communis (XM_002510439.1)
SEQ ID NO: 136 полинуклеотид CGi58 Medicago truncatula (XM_003603685.1)
SEQ ID NO: 137 полинуклеотид LEC2 Arabidopsis thaliana (NM_102595.2)
SEQ ID NO: 138 полинуклеотид LEC2 Medicago truncatula (X60387.1)
SEQ ID NO: 139 полинуклеотид LEC2 Brassica napus (HM370539.1)
SEQ ID NO: 140 полинуклеотид BBM Arabidopsis thaliana (NM_121749.2)
SEQ ID NO: 141 полинуклеотид BBM Medicago truncatula (AY899909.1)
SEQ ID NO: 142 полипептид LEC2 Arabidopsis thaliana (NP_564304.1)
SEQ ID NO: 143 полипептид LEC2 Medicago truncatula (CAA42938.1)
SEQ ID NO: 144 полипептид LEC2 Brassica napus (ADO16343.1)
SEQ ID NO: 145 полипептид BBM Arabidopsis thaliana (NP_197245.2)
SEQ ID NO: 146 полипептид BBM Medicago truncatula (AAW82334.1)
SEQ ID NO: 147 индуцибельный промотор alcA Aspergilus niger
SEQ ID NO: 148 индуктор AlcR, который активирует промотор AlcA в присутствии этанола
SEQ ID NO: 149 LEC1 Arabidopsis thaliana (AAC39488)
SEQ ID NO: 150 LEC1 Arabidopsis lyrata (XP_002862657)
SEQ ID NO: 151 LEC1 Brassica napus (ADF81045)
SEQ ID NO: 152 LEC1 Ricinus communis (XP_002522740)
SEQ ID NO: 153 LEC1 Glycine max (XP_006582823)
SEQ ID NO: 154 LEC1 Medicago truncatula (AFK49653)
SEQ ID NO: 155 LEC1 Zea mays (AAK95562)
SEQ ID NO: 156 LEC1 Arachis hypogaea (ADC33213)
SEQ ID NO: 157 LEC1-подобного Arabidopsis thaliana (AAN15924)
SEQ ID NO: 158 LEC1-подобного Brassica napus (AHI94922)
SEQ ID NO: 159 LEC1-подобного Phaseolus coccineus (AAN01148)
SEQ ID NO: 160 FUS3 Arabidopsis thaliana (AAC35247)
SEQ ID NO: 161 FUS3 Brassica napus
SEQ ID NO: 162 FUS3 Medicago truncatula
SEQ ID NO: 163 последовательность кДНК SDP1 Arabidopsis thaliana, номер доступа NM_120486, 3275 нуклеотидов (нт)
SEQ ID NO: 164 кДНК SDP1 Brassica napus номер доступа GN078290
SEQ ID NO: 165 кДНК SDP1 Brachypodium distachyon, 2670 нуклеотидов (нт)
SEQ ID NO: 166 кДНК SDP1 Populus trichocarpa, 3884 нт
SEQ ID NO: 167 кДНК SDP1 Medicago truncatula; XM_003591377; 2490 нт
SEQ ID NO: 168 кДНК SDP1Glycine max XM_003521103; 2783 нт
SEQ ID NO: 169 кДНК SDP1Sorghum bicolor XM_002458486; 2724 нт
SEQ ID NO: 170 кДНК SDP1Zea mays, NM_001175206; 2985 нт
SEQ ID NO: 171 кДНК SDP1Physcomitrella patens, XM_001758117; 1998 нт
SEQ ID NO: 172 кДНК SDP1 Hordeum vulgare, AK372092; 3439 нт
SEQ ID NO: 173 кДНК SDP1Nicotiana benthamiana, Nbv5tr6404201
SEQ ID NO: 174 участок кДНК SDP1 Nicotiana benthamiana, являющийся мишенью для сайленсинга шпРНКи
SEQ ID NO: 175 промотор гена SDP1 Arabidopsis thaliana, 1,5 тысяч пар оснований
SEQ ID NO: 176 нуклеотидная последовательность, комплементарная гену pSSU-Олеозина в Т-ДНК pJP3502. В порядке (комплементарной последовательности): терминатор лектина Glycine max 348 нт, 3'-экзон 255 нт, интрон UBQ10 304 нт, 5'-экзон 213 нт, промотор SSU 1751 нт
SEQ ID NO: 177 кДНК пластидной GPAT Arabidopsis thaliana, NM_179407
SEQ ID NO: 178 полипептид пластидной GPAT Arabidopsis thaliana, NM_179407
SEQ ID NO: 179 кДНК пластидной GPAT Populus trichocarpa, XP_006368351
SEQ ID NO: 180 кДНК пластидной GPAT Jatropha curcas, ACR61638
SEQ ID NO: 181 кДНК пластидной GPAT Ricinus communis, XP_002518993
SEQ ID NO: 182 кДНК пластидной GPAT Helianthus annuus, ADV16382
SEQ ID NO: 183 кДНК пластидной GPAT Medicago truncatula, XP_003612801
SEQ ID NO: 184 кДНК пластидной GPAT Glycine max, XP_003516958
SEQ ID NO: 185 кДНК пластидной GPAT Carthamus tinctorius, CAHG3PACTR
SEQ ID NO: 186 кДНК пластидной GPAT Solanum tuberosum, XP_006352898
SEQ ID NO: 187 кДНК пластидной GPAT Oryza sativa, японской, NM_001072027
SEQ ID NO: 188 кДНК пластидной GPAT Sorghum bicolor, XM_002467381
SEQ ID NO: 189 кДНК пластидной GPAT Zea mays, NM_001158637
SEQ ID NO: 190 кДНК пластидной GPAT Hordeum vulgare, AK371419
SEQ ID NO: 191 кДНК пластидной GPAT Physcomitrella patens, XM_001771247
SEQ ID NO: 192 кДНК пластидной GPAT Chlamydomonas reinhardtii, XM_001694925
SEQ ID NO: 193 FATA1 Arabidopsis thaliana
SEQ ID NO: 194 FATA2 Arabidopsis thaliana
SEQ ID NO: 195 FATB Arabidopsis thaliana
SEQ ID NO: 196 WRI3 Arabidopsis thaliana
SEQ ID NO: 197 WRI4 Arabidopsis thaliana
SEQ ID NO: 198 WRI1 Avena sativa
SEQ ID NO: 199 WRI1 Sorghum bicolor
SEQ ID NO: 200 WRI1 Zea mays
SEQ ID NO: 201 WRI1 Triadica sebifera
SEQ ID NO: 202 последовательность промотора пататина B33 S. tuberosum
SEQ ID NO 203-206 и 236-245 олигонуклеотидные праймеры
SEQ ID NO: 207 промоторная область SEE1 Z. mays (1970 нт из номера доступа AJ494982)
SEQ ID NO: 208 последовательность промотора AlSAP A. littoralis, номер доступа DQ885219
SEQ ID NO: 209 последовательность промотора ArRolC A. rhizogenes, номер доступа DQ160187
SEQ ID NO: 210 конструкция шпРНКи, содержащая фрагмент 732 пары оснований пластидной GPAT N. benthamiana
SEQ ID NO: 211 DGAT1 Elaeis guineensis (масличная пальма)
SEQ ID NO: 212 MYB73 G. max, номер доступа ABH02868
SEQ ID NO: 213 bZIP53 A. thaliana, номер доступа AAM14360
SEQ ID NO: 214 AGL15 A. thaliana, номер доступа NP_196883
SEQ ID NO: 215 MYB118 A. thaliana, номер доступа AAS58517
SEQ ID NO: 216 MYB115 A. thaliana, номер доступа AAS10103
SEQ ID NO: 217 TANMEI A. thaliana, номер доступа BAE44475
SEQ ID NO: 218 WUS A. thaliana, номер доступа NP_565429
SEQ ID NO: 219 GFR2a1 B. napus, номер доступа AFB74090
SEQ ID NO: 220 GFR2a2 B. napus, номер доступа AFB74089
SEQ ID NO: 221 PHR1 A. thaliana, номер доступа AAN72198
SEQ ID NO: 222 фрагмент TGD1 N. benthamiana
SEQ ID NO: 223 аминокислоты SDP1 картофеля
SEQ ID NO: 224 нуклеотидная последовательность SDP1 картофеля
SEQ ID NO: 225 малая субъединица АГФ-азы картофеля
SEQ ID NO: 226 нуклеотидная последовательность малой субъединицы АГФ-азы картофеля
SEQ ID NO: 227 нуклеотидная последовательность LDAP-1 Sapium sebiferum
SEQ ID NO: 228 аминокислотная последовательность LDAP-1Sapium sebiferum
SEQ ID NO: 229 нуклеотидная последовательность LDAP-2 Sapium sebiferum
SEQ ID NO: 230 аминокислотная последовательность LDAP-2 Sapium sebiferum
SEQ ID NO: 231 нуклеотидная последовательность LDAP-3 Sapium sebiferum
SEQ ID NO: 232 аминокислотная последовательность LDAP-3 Sapium sebiferum
SEQ ID NO: 233 SDP1 S. bicolor (номер доступа XM_002463620)
SEQ ID NO: 234 нуклеотидная последовательность SDP1 T. aestivum (номер доступа AK334547)
SEQ ID NO: 235 фрагмент шпРНКи SDP1 S. bicolor
SEQ ID NO:246 последовательность промотора DIRIGENT (DIR16) гибрида Saccharum
SEQ ID NO:247 последовательность промотора O-метил-трансферазы (OMT) гибрида Saccharum
SEQ ID NO:248 Последовательность аллеля A1 промотора гена R1MYB1 гибрида Saccharum
SEQ ID NO:249 последовательность промотора гена нагрузки стебля 5 (LSG5) гибрида Saccharum Loading Stem Gene
SEQ ID NO:250 Нуклеотидная последовательность кодирующей белок области кДНК гена TGD5 Sorghum bicolor, номер доступа XM_002442154; 297 нт
SEQ ID NO:251 Аминокислотная последовательность полипептида TGD5 Sorghum bicolor, номер доступа XM_002442154; 98 ак
SEQ ID NO:252 Нуклеотидная последовательность кодирующей белок области кДНК гена TGD5 Zea mays, номер доступа EU972796.1; 297 нт
SEQ ID NO:253 Аминокислотная последовательность полипептида TGD5 Zea mays, номер доступа EU972796.1; 98 ак
SEQ ID NO:254 Нуклеотидная последовательность кодирующей белок области кДНК гена, кодирующего малую субъединицу АГФазы Sorghum bicolor (номер доступа XM_002462095.1); 1533 нт
SEQ ID NO:255 Аминокислотная последовательность полипептида малой субъединицы АГФазы Sorghum bicolor (номер доступа XM_002462095.1); 510 ак
SEQ ID NO:256 Нуклеотидная последовательность кодирующей белок области кДНК гена, кодирующего полипептид малой субъединицы АГФазы Zea mays (номер доступа XM_008666513.1); 1554 нт
SEQ ID NO:257 Аминокислотная последовательность полипептида малой субъединицы АГФазы Zea mays (номер доступа XM_008666513.1); 517 ак
SEQ ID NO:258 Нуклеотидная последовательность кодирующей белок области кДНК гена PDAT1 Sorghum bicolor (номер доступа XM_002462417.1);
SEQ ID NO:259 Аминокислотная последовательность полипептида PDAT1 Sorghum bicolor (номер доступа XM_002462417.1); 682 ак
SEQ ID NO:260 Нуклеотидная последовательность кодирующей белок области кДНК гена PDAT1 Zea mays (номер доступа NM_001147943); 2037 нт
SEQ ID NO:261 Аминокислотная последовательность полипептида Zea mays PDAT1 (номер доступа NM_001147943); 678 ак
SEQ ID NO:262 Нуклеотидная последовательность кодирующей белок области кДНК гена PDCT Sorghum bicolor (номер доступа XM_002437214); 846 нт
SEQ ID NO:263 Аминокислотная последовательность полипептида PDCT Sorghum bicolor (номер доступа XM_002437214); 281 ак
SEQ ID NO:264 Нуклеотидная последовательность кодирующей белок области кДНК гена PDCT Zea mays (номер доступа EU973573.1); 849 нт
SEQ ID NO:265 Аминокислотная последовательность полипептида PDCT Zea mays (номер доступа EU973573.1); 282 ак
SEQ ID NO:266 Нуклеотидная последовательность кодирующей белок области кДНК гена TST1 Sorghum bicolor (номер доступа XM_002467535.1); 2223 нт
SEQ ID NO:267 Аминокислотная последовательность полипептида TST1 Sorghum bicolor (номер доступа XM_002467535.1); 740 ак
SEQ ID NO:268 Нуклеотидная последовательность кодирующей белок области кДНК гена TST1 Zea mays (номер доступа NM_001158464); 2244 нт
SEQ ID NO:269 Аминокислотная последовательность полипептида TST1 Zea mays (номер доступа NM_001158464); 747 ак
SEQ ID NO:270 Нуклеотидная последовательность кодирующей белок области кДНК гена TST2 Sorghum bicolor (Sb04G008150; Sobic.004G099300; номер доступа KXG29849.1); 2238 нт
SEQ ID NO:271 Аминокислотная последовательность полипептида TST2 Sorghum bicolor (номер доступа KXG29849.1); 745 ак
SEQ ID NO:272 Нуклеотидная последовательность кодирующей белок области кДНК гена TST2 Zea mays (номер доступа XM_008647398.1); 2238 нт
SEQ ID NO:273 Аминокислотная последовательность полипептида TST2 Zea mays (номер доступа XM_008647398.1); 745 ак
SEQ ID NO:274 Нуклеотидная последовательность кодирующей белок области кДНК гена INV3 Sorghum bicolor (Sobic.004G004800; Sb04g000620; номер доступа XM_002451312); 1464 нт
SEQ ID NO:275 Аминокислотная последовательность полипептида INV3 Sorghum bicolor (номер доступа XM_002451312); 487 ак
SEQ ID NO:276 Аминокислотная последовательность полипептида INV3 Sorghum bicolor; более длинная форма альтернативного сплайсинга (номер доступа EES04332,2); 638 ак
SEQ ID NO:277 Нуклеотидная последовательность кодирующей белок области кДНК гена INV2 Zea mays (гомолог маиса для INV3 Sb) (номер доступа NM_001305860.1); 2022 нт
SEQ ID NO:278 Аминокислотная последовательность полипептида INV2 Zea mays (гомолог маиса для INV3 Sb) (номер доступа NM_001305860.1); 673 ак
SEQ ID NO:279 Нуклеотидная последовательность кодирующей белок области кДНК гена SUS4 Sorghum bicolor (Sobic.001G344500; Sb01g033060; номер доступа XM_002465116.1); 2451 нт
SEQ ID NO:280 Аминокислотная последовательность полипептида SUS4 Sorghum bicolor (номер доступа XM_002465116.1); 816 ак
SEQ ID NO:281 Нуклеотидная последовательность кодирующей белок области кДНК гена SUS1 Zea mays gene (гомолог маиса для SUS4 Sb) (номер доступа NM_001111853); 2451 нт
SEQ ID NO:282 Аминокислотная последовательность полипептида SUS1 Zea mays (номер доступа NM_001111853); 816 ак
SEQ ID NO:283 Нуклеотидная последовательность кодирующей белок области кДНК гена bCIN Sorghum bicolor (Sobic.004G172700; Sb04g022350; номер доступа XM_002453920.1);
SEQ ID NO:284 Аминокислотная последовательность полипептида bCIN Sorghum bicolor (номер доступа XM_002453920.1); 559 ак
SEQ ID NO:285 Нуклеотидная последовательность кодирующей белок области кДНК гена цитозольного INV Zea mays (гомолог для bCIN Sb) (номер доступа NM_001175248.1); 1680 нт
SEQ ID NO:286 Аминокислотная последовательность полипептида INV Zea mays (номер доступа NM_001175248,1); 559 ак
SEQ ID NO:287 Нуклеотидная последовательность кодирующей белок области кДНК гена SUT4 Sorghum bicolor (Sb04g038030; номер доступа XM_002453038.1); 1785 нт
SEQ ID NO:288 Аминокислотная последовательность полипептида SUT4 Sorghum bicolor (номер доступа XM_002453038.1); 594 ак
SEQ ID NO:289 Нуклеотидная последовательность кодирующей белок области кДНК гена SUT2 Zea mays (номер доступа AY581895.1); 1779 нт
SEQ ID NO:290 Аминокислотная последовательность полипептида SUT2 Zea mays (номер доступа AY581895.1); 592 ак
SEQ ID NO:291 Нуклеотидная последовательность кодирующей белок области кДНК гена SWEET16 Arabidopsis thaliana (номер доступа NM_001338249.1); 693 нт
SEQ ID NO:292 Аминокислотная последовательность полипептида SWEET16 Arabidopsis thaliana (номер доступа NM_001338249.1); 230 ак
SEQ ID NO:293 Нуклеотидная последовательность кодирующей белок области кДНК гена MED15-1 Arabidopsis thaliana (номер доступа NM_101446.4); 4008 нт
SEQ ID NO:294 Аминокислотная последовательность полипептида MED15-1 Arabidopsis thaliana (номер доступа NM_101446.4); 1335 ак
SEQ ID NO:295 Нуклеотидная последовательность кодирующей белок области кДНК гена MED15-1 Zea mays (номер доступа NM_001321633.1); 3927 нт
SEQ ID NO:296 Аминокислотная последовательность полипептида MED15-1 Zea mays (номер доступа NM_001321633.1); 1308 ак
SEQ ID NO:297 Нуклеотидная последовательность кодирующей белок области кДНК гена 14-3-3 Arabidopsis thaliana (номер доступа AY079350);
SEQ ID NO:298 Аминокислотная последовательность полипептида 14-3-3 Arabidopsis thaliana (номер доступа AY079350); 248 ак
SEQ ID NO:299 Нуклеотидная последовательность кодирующей белок области кДНК гена 14-3-3 Sorghum bicolor (номер доступа XM_002445734.1); 762 нт
SEQ ID NO:300 Аминокислотная последовательность полипептида 14-3-3 Sorghum bicolor (номер доступа XM_002445734.1); 253 ак
SEQ ID NO:301 Нуклеотидная последовательность кодирующей белок области кДНК гена 14-3-3 Arabidopsis thaliana (номер доступа NM_001203346); 777 нт
SEQ ID NO:302 Аминокислотная последовательность полипептида 14-3-3 Arabidopsis thaliana (номер доступа NM_001203346); 258 ак
SEQ ID NO:303 Нуклеотидная последовательность кодирующей белок области кДНК гена 14-3-3 Sorghum bicolor (номер доступа XM_002445734.1); 762 нт
SEQ ID NO:304 Аминокислотная последовательность полипептида 14-3-3 Sorghum bicolor (номер доступа XM_002445734.1); 253 ак
SEQ ID NO:305 Аминокислотная последовательность полипептида олеозина L Sesamum indicum (номер доступа AF091840)
SEQ ID NO:306 Аминокислотная последовательность полипептида ортолога олеозина L Ficus pumila var. awkeotsang (номер доступа ABQ57397.1)
SEQ ID NO:307 Аминокислотная последовательность полипептида ортолога олеозина L Cucumis sativus (номер доступа XP_004146901.1)
SEQ ID NO:308 Аминокислотная последовательность полипептида ортолога олеозина L Linum usitatissimum (номер доступа ABB01618.1)
SEQ ID NO:309 Аминокислотная последовательность полипептида ортолога олеозина L Glycine max (номер доступа XP_003556321.2)
SEQ ID NO:310 Аминокислотная последовательность полипептида ортолога олеозина L Ananas comosus (номер доступа OAY72596.1)
SEQ ID NO:311 Аминокислотная последовательность полипептида ортолога олеозина L Setaria italica (номер доступа XP_004956407.1)
SEQ ID NO:312 Аминокислотная последовательность полипептида ортолога олеозина L Fragaria vesca подвид vesca (номер доступа XP_004307777.1)
SEQ ID NO:313 Аминокислотная последовательность полипептида ортолога олеозина L Brassica napus (номер доступа CDY03377.1)
SEQ ID NO:314 Аминокислотная последовательность полипептида ортолога олеозина L Solanum lycopersicum (номер доступа XP_004240765.1)
SEQ ID NO: 315. Аминокислотная последовательность олеозина U1 Vanilla planifolia
SEQ ID NO: 316. Аминокислотная последовательность TsLDAP1 Triadica sebifera (китайское сальное дерево)
SEQ ID NO: 317. Аминокислотная последовательность TsLDAP2 Triadica sebifera (китайское сальное дерево)
SEQ ID NO: 318. Аминокислотная последовательность TsLDAP3 Triadica sebifera (китайское сальное дерево)
SEQ ID NO: 319. Аминокислотная последовательность GPAT9 Cocos nucifera (кокос)
SEQ ID NO: 320. Аминокислотная последовательность CPT1 Zea mays (номер доступа NP_001151915.1)
SEQ ID NO: 321. Аминокислотная последовательность CPT1 Zea mays (номер доступа XP_008649199.1)
SEQ ID NO: 322. Аминокислотная последовательность CPT1 Sorghum bicolor (номер доступа XP_002451408.1)
SEQ ID NO: 323. Аминокислотная последовательность CPT1 Sorghum bicolor (номер доступа XP_021305900.1)
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Общие способы
Если конкретно не определено иное, все технические и научные термины, используемые в настоящем описании, следует интерпретировать как имеющие такое же значение, которое им обычно придает специалист в данной области (например, культуры клеток, молекулярной генетики, биологии растений, клеточной биологии, химии белка, химии липида и жирной кислоты, питания животных, получения биотоплива и биохимии).
Если не указано иное, способы химии рекомбинантного белка, культуры клетки и иммунологические способы, применяемые в настоящем изобретении, представляют собой стандартные способы, хорошо известные специалистам из уровня техники. Такие способы описаны и объяснены в литературных источниках, например, J. Perbal, A Practical Guide to Molecular Cloning, John Wiley and Sons (1984), J. Sambrook et al., Molecular Cloning: A Laboratory Manual, Cold Spring Harbour Laboratory Press (1989), T.A. Brown (редактор), Essential Molecular Biology: A Practical Approach, Volumes 1 and 2, IRL Press (1991), D.M. Glover and B.D. Glover and B.D. Hames (редакторы), DNA Cloning: A Practical Approach, Volumes 1-4, IRL Press (1995 и 1996), F.M. Ausubel et al. (редакторы), Current Protocols in Molecular Biology, Greene Pub. Associates and Wiley-Interscience (1988, включая все обновления, изданные до настоящего времени), Ed Harlow and David Lane (редакторы) Antibodies: A Laboratory Manual, Cold Spring Harbour Laboratory, (1988), и J.E. Coligan et al. (редакторы), Current Protocols in Immunology, John Wiley & Sons (включая все обновления, изданные до настоящего времени).
Некоторые определения
Термин «экзогенный» в контексте полинуклеотида или полипептида обозначает полинуклеотид или полипептид, присутствующий в клетке или растении, или его части, которые от природы не содержат полинуклеотида или полипептида. Такая клетка в настоящем описании названа «рекомбинантной клеткой» или «трансгенной клеткой», и растение, содержащее клетку - «трансгенным растением». В одном варианте осуществления, экзогенный полинуклеотид или полипептид происходит из другого рода относительно клетки или растения, или его части,, содержащих экзогенный полинуклеотид или полипептид. В другом варианте осуществления экзогенный полинуклеотид или полипептид происходит от другого вида. В одном варианте осуществления экзогенный полинуклеотид или полипептид, экспрессированный в клетке растения, происходит от другого вида или рода. Экзогенный полинуклеотид или полипептид может быть неприродным, например, таким как синтетическая молекула ДНК, которая получена способами рекомбинации ДНК. Молекула ДНК может, предпочтительно, содержать кодирующий белок участок, который является кодон-оптимизированным для экспрессии в клетке растения, с получением таким образом полипептида, который содержит такую же последовательность аминокислот, что и природный полипептид, даже если нуклеотидная последовательность кодирующего белок участка имеет неприродное происхождение. Экзогенный полинуклеотид может кодировать или экзогенный полипептид может, например, представлять собой: диацилглицерин-ацилтрансферазу (DGAT), например, DGAT1 или DGAT2, фактор транскрипции Wrinkled 1 (WRI1), на OBC, таком как олеозин или, предпочтительно, LDAP, тиоэстераза жирной кислоты, такая как полипептид FATA или FATB или полипептид супрессора сайленсинга. В одном варианте осуществления, клетка по изобретению представляет собой рекомбинантную клетку.
Как используют в настоящем описании, термин «содержание триацилглицерина (TAG)» или его варианты относится к количеству TAG в клетке, растении или его части. Содержание TAG можно рассчитывать с использованием способов, известны в данной области, как суммарное содержание групп глицерина и жирного ацила с использованием соотношения: % TAG по массе=100x ((41x общее количество моль FAME/3)+( общее количсество г FAME - (15x общее количество моль FAME)))/г, где 41 и 15 представляют собой молекулярные массы группы глицерина и метильной группы, соответственно (где FAME представляет собой сложные метильные эфиры жирных кислот) (см. Примеры, такие как Пример 1).
Как используют в настоящем описании, термин «общее содержание жирных кислот (TFA)» или его варианты относится к общему количеству жирных кислот в клетке, растении или его части в пересчете на массу, как процент от массы клетки, растения или его части. Если не указано иное, масса клетки, растения или его части представляет собой сухую массу клетки, растения или его части. Содержание TFA измеряют, как описано в Примере 1 в настоящем описании. Способ включает перевод жирных кислот в образце в FAME и измерение количества FAME посредством GC, с использованием добавления известного количества отличительного стандарта жирных кислот, такого как C17:0, в качестве количественного стандарта в GC. TFA, таким образом, представляет массу только жирных кислот, не массу жирных кислот и связанных с ними групп в липиде растения.
Как используют в настоящем описании, параметр «соотношение TAG/TFA» или «TTQ» рассчитывают как уровень TAG (%), деленный на уровень TFA (%), каждый как процент от сухой массы материала растения. Например, уровень TAG 6%, содержащийся в уровне TFA 10%, приводит к TTQ 0,6. Уровни TAG и TFA измеряют, как описано в настоящем описании. Понятно, что, в этом контексте, уровень TFA относится к массе общего содержания жирных кислот, и уровень TAG относится к массе TAG, включая глицериновую группу TAG.
Как используют в настоящем описании, термин «содержание растворимого белка» или его варианты относится к количеству растворимого белка в растении или его части. Содержание растворимого белка можно рассчитывать с использованием способов, известных в данной области. Например, свежую ткань можно измельчать, хлорофилл и растворимые сахара экстрагировать посредством нагревания до 80°C в 50-80% (об./об.) этаноле в 2,5 мМ буфере HEPES при pH 7,5, центрифурирования, промывки осадка в дистиллированной воде, ресуспендирования осадка в 0,1 M NaOH и нагревания до 95°C в течение 30 мин, и затем использования анализа Брэдфорд (Bradford, 1976), определяющего содержание растворимого белка. Альтернативно, свежую ткань можно измельчать в буфере, содержащем 100 мМ Tris-HCl pH 8,0 и 10 мМ MgCl2.
Как используют в настоящем описании, термин «содержание азота» или его варианты относится к количеству азота в растении или его части. Содержание азота можно рассчитывать с использованием способов, известных в данной области. Например, лиофилизированную ткань можно анализировать с использованием масс-спектрометра изотопных соотношений с системой подготовки ANCA, содержащей трубку для сжигания и восстановления, функционирующую при 1000°C и 600°C, соответственно, для определения содержания азота.
Как используют в настоящем описании, термин «содержание углерода» или его варианты относится к количеству углерода в растении или его части. Содержание углерода можно рассчитывать с использованием способов, известных в данной области. Например, уровни органического углерода можно определять с использованием способа, описанного в Shaw (1959), или как описано в Примере 1.
Как используют в настоящем описании, термин «соотношение углерод:азот» или или его варианты относится к относительному количеству углерода в клетке, растении или его части по сравнению с количеством азота в клетке, растении или его части. Содержание углерода и азота можно рассчитывать, как описано выше, и представлять в форме соотношения.
Как используют в настоящем описании, термин «экспрессия фотосинтетического гена» или его варианты относится к одному или нескольким генам, экспрессирующих белки, вовлеченные в фотосинтетические пути в растении или его части. Примеры фотосинтетических генов, которые могут подвергаться повышающей регуляции в растениях или их частях по изобретению, включают, но без ограничения, один или несколько из генов, перечисленных в Таблице 10.
Как используют в настоящем описании, термин «способность к фотосинтезу» или его варианты относится к способности растения или его части осуществлять фотосинтез (переводить энергию света в химическую энергию). Способность к фотосинтезу (Amax) представляет собой показатель максимальной скорости, с которой листья способны фиксировать углерод в ходе фотосинтеза. Ее, как правило, измеряют как количество диоксида углерода, фиксируемого на квадратный метр в секунду, например, как мкмоль м-2 с-1. Способность к фотосинтезу можно рассчитывать с использованием способов, известных в данной области.
Как используют в настоящем описании, термин «общее содержание пищевых волокон (TDF)» или его варианты относится к количеству волокон (включая растворимые и нерастворимые волокна) в клетке, растении или его части. Как понятно специалисту в данной области, пищевые волокна включают некрахмальные полисахариды, такие как арбиноксиланы, целлюлоза, и многие другие компоненты растений, такие как недеградируемый крахмал, недеградируемые декстрины, инулин, лигнин, хитины, пектины, β-глюканы и олигосахариды. TDF можно рассчитывать с использованием способов, известных в данной области. Например, с использованием способа Prosky (Prosky et al. 1985), способа McCleary (McCleary et al., 2007) или быстрый комплексный способ определения общего содержания пищевых волокон (McCleary et al., 2015).
Как используют в настоящем описании, термин «содержание энергии» или его варианты относится к количеству пищевой энергии в растении или его части. Более конкретно, количество химической энергии, которое животные (включая человека) получают из пищи. Содержание энергии можно рассчитывать с использованием способов, известных в данной области. Например, содержание энергии можно определять на основании теплоты сгорания в бомбовом калориметре и коррекций, принимающих во внимание эффективность переваривания и всасывания, и продукцию мочевины и других веществ в моче. В качестве другого примера, содержание энергии можно рассчитывать, как описано в Примере 1.
В настоящем описании термин «экстрагированный липид» обозначает состав, экстрагированный из клетки, растения или его части по изобретению, таких как трансгенные клетка, растение или его часть по изобретению, содержащие по меньшей мере 60% (масс./масс.) липида.
В настоящем описании, термин «неполярный липид» обозначает жирные кислоты и их производные, растворимые в органических растворителях, но не растворимые в воде. Жирные кислоты могут представлять собой жирные кислоты в свободной и/или этерифицированной форме. Примеры этерифицированных форм неполярного липида включают, но без ограничения, триацилглицерин (TAG), диацилглицерин (DAG), моноацилглицерин (MAG). Неполярные липиды также включают стеролы, эфиры стеролов и эфиры восков. Неполярные липиды также известны, как «нейтральные липиды». При комнатной температуре неполярный липид обычно представляет собой жидкость. Предпочтительно, неполярный липид в основном (> 50%) содержит жирные кислоты, длина которых составляет по меньшей мере 16 атомов углерода. Более предпочтительно, по меньшей мере 50% общих жирных кислот в неполярном липиде представляют собой C18 жирные кислоты, например, олеиновую кислоту. В одном варианте осуществления, по меньшей мере 5% от общих жирных кислот в неполярных липидах представляют собой жирные кислоты C12 или C14 или те и другие. В одном варианте осуществления, по меньшей мере 50%, более предпочтительно, по меньшей мере 70%, более предпочтительно, по меньшей мере 80%, более предпочтительно, по меньшей мере 90%, более предпочтительно, по меньшей мере 91%, более предпочтительно, по меньшей мере 92%, более предпочтительно, по меньшей мере 93%, более предпочтительно, по меньшей мере 94%, более предпочтительно, по меньшей мере 95%, более предпочтительно, по меньшей мере 96%, более предпочтительно, по меньшей мере 97%, более предпочтительно, по меньшей мере 98%, более предпочтительно, по меньшей мере 99% жирных кислот в неполярном липиде по изобретению присутствуют в форме TAG. Неполярный липид может быть дополнительно очищен или обработан, например, посредством гидролиза с использованием сильного основания, чтобы высвободить свободную жирную кислоту, или посредством фракционирования, дистилляции или т.п. Неполярный липид может присутствовать в частях растений, например, семени, листьях, клубнях, свекле или плоде, или быть получен из них. Неполярный липид по изобретению может образовывать часть «масла из семян», если он получен из семени.
Концентрация свободного и этерифицированного стерола (например, ситостерола, кампэстрола, стигмастерола, брассикастерола, Δ5-авенастерола, ситостанола, кампестанола и холестерина) в экстрагированном липиде может быть такой, как описано в Phillips et al. (2002). Стеролы в растительных маслах присутствуют, как свободные спирты, эфиры с жирными кислотами (этерифицированные стеролы), гликозиды стеролов и ацилированные гликозиды стеролов. Концентрации стеролов в природных растительных маслах (масло из семян) варьируют до максимального содержания приблизительно 1100 мг/100 г. Гидрогенизированное пальмовое масло содержит одну из самых низких концентраций для природных масел, приблизительно 60 мг/100 г. Извлеченные или экстрагированные масла из семян по изобретению предпочтительно, содержат от приблизительно 100 до приблизительно 1000 мг общего стерола/100 г масла. В случае применения в качестве продукта питания или корма, предпочтительно, стеролы присутствуют, главным образом, в свободных или этерифицированных формах вместо гликозилированных форм. В маслах из семян по настоящему изобретению, предпочтительно, по меньшей мере 50% стеролов в маслах присутствуют, как этерифицированные стеролы, за исключением соевого масла, которое содержит приблизительно 25% этерифицированных стеролов. Масло из семени рапса и рапса по изобретению предпочтительно, содержат от приблизительно 500 до приблизительно 800 мг общего стерола/100 г, где основным стеролом является ситостерол, и вторым по распространенности - кампэстрол. Кукурузное масло из семени по изобретению предпочтительно, содержит от приблизительно 600 до приблизительно 800 мг общего стерола/100 г, где основным стеролом является ситостерол. Соевое масло из семени по изобретению предпочтительно, содержит от приблизительно 150 до приблизительно 350 мг общего стерола/100 г, где основным стеролом является ситостерол, и вторым по распространенности - стигмастерол, где содержание свободного стерола выше, чем этерицифированного стерола. Хлопковое масло по изобретению предпочтительно, содержит от приблизительно 200 до приблизительно 350 мг общего стерола/100 г, где основным стеролом является ситостерол. Кокосовое масло и пальмовое масло по изобретению предпочтительно, содержат от приблизительно 50 до приблизительно 100 мг общего стерола/100 г, где основным стеролом является ситостерол. Сафлоровое масло из семени по изобретению предпочтительно, содержит от приблизительно 150 до приблизительно 250 мг общего стерола/100 г, где основным стеролом является ситостерол. Арахисовое масло по изобретению предпочтительно, содержит от приблизительно 100 до приблизительно 200 мг общего стерола/100 г, где основным стеролом является ситостерол. Кунжутное масло из семени по изобретению предпочтительно, содержит от приблизительно 400 до приблизительно 600 мг общего стерола/100 г, где основным стеролом является ситостерол. Масло подсолнечника из семени по изобретению предпочтительно, содержит от приблизительно 200 до 400 мг общего стерола/100 г, где основным стеролом является ситостерол. Масла, полученные из вегетативных частей растений по изобретению, предпочтительно, содержат менее 200 мг общего стерола/100 г, более предпочтительно, менее 100 мг общего стерола/100 г, и наиболее предпочтительно, менее 50 мг общего стерола/100 г, где большая часть стеролов присутствует в форме свободных стеролов.
В настоящем описании, термин «растительное масло» обозначает состав, полученный из вегетативных частей растения, содержащий по меньшей мере 60% (масс./масс.) липидов, или такой, который можно получить из вегетативных частей, если масло все еще присутствует в вегетативной части. Таким образом, растительное масло по изобретению включает масло, которое присутствует в вегетативной части растения, а также масло, которое экстрагировано из вегетативной части (экстрагированное масло). Растительное масло, предпочтительно, представляет собой экстрагированное растительное масло. Растительное масло при комнатной температуре обычно представляет собой жидкость. Предпочтительно, общее содержание жирных кислот (TFA) в растительном масле в основном (> 50%) включает жирные кислоты, длина которых составляет по меньшей мере 16 атомов углерода. Более предпочтительно, по меньшей мере 50% общих жирных кислот в растительном масле представляют собой C18 жирные кислоты например, олеиновую кислоту. Жирные кислоты обычно присутствуют в этерифицированной форме, например, такой как TAG, DAG, ацил-CoA, галактолипид или фосфолипид. Жирные кислоты могут быть жирными кислотами в свободной и/или этерифицированной форме. В одном варианте осуществления, по меньшей мере 50%, более предпочтительно, по меньшей мере 70%, более предпочтительно, по меньшей мере 80%, более предпочтительно, по меньшей мере 90%, более предпочтительно, по меньшей мере 91%, более предпочтительно, по меньшей мере 92%, более предпочтительно, по меньшей мере 93%, более предпочтительно, по меньшей мере 94%, более предпочтительно, по меньшей мере 95%, более предпочтительно, по меньшей мере 96%, более предпочтительно, по меньшей мере 97%, более предпочтительно, по меньшей мере 98%, более предпочтительно, по меньшей мере 99% жирных кислот в растительном масле по изобретению могут быть обнаружены в виде TAG. В одном варианте осуществления, растительное масло по изобретению представляет собой «в основном очищенное» или «очищенное» масло, которое отделено от одного или нескольких других липидов, нуклеиновых кислот, полипептидов или других загрязняющих молекул, с которыми оно ассоциировано в вегетативной части растения или в неочищенном экстракте. Предпочтительно, в основном очищенное растительное масло по меньшей мере на 60% свободно, более предпочтительно, по меньшей мере на 75% свободно, и более предпочтительно, по меньшей мере на 90% свободно от других компонентов, с которыми оно ассоциировано в семени или экстракте. Растительное масло по изобретению может дополнительно содержать молекулы, не относящиеся к жирным кислотам, например, но без ограничения, стеролы. В одном варианте осуществления, растительное масло представляет собой масло рапса (вида Brassica, такой как Brassica carinata, Brassica juncea, Brassica napobrassica, Brassica napus), горчичное масло (Brassica juncea), масло других видов Brassica (например, Brassica napobrassica, Brassica camelina), масло подсолнечника (вида Helianthus, такой как Helianthus annuus), льняное масло (Linum usitatissimum), соевое масло (Glycine max), сафлоровое масло (Carthamus tinctorius), кукурузное масло (Zea mays), масло табака (вида Nicotiana, такой как Nicotiana tabacum или Nicotiana benthamiana), арахисовое масло (Arachis hypogaea), пальмовое масло (Elaeis guineensis), хлопковое масло (Gossypium hirsutum), кокосовое масло (Cocos nucifera), масло авокадо (Persea americana), оливковое масло (Olea europaea), масло кешью (Anacardium occidentale), масло макадамии (Macadamia intergrifolia), миндальное масло (Prunus amygdalus), овсяное масло (Avena sativa), рисовое масло (вида Oryza, такой как Oryza sativa и Oryza glaberrima), масло из Arabidopsis (Arabidopsis thaliana), Aracinis hypogaea (арахис), Beta vulgaris (сахарная свекла), Camelina sativa (ложный лен), Crambe abyssinica (абиссинская капуста), Cucumis melo (дыня), Hordeum vulgare (ячмень), Jatropha curcas (лечебный орех), Joannesia princeps (лещина арара), Licania rigida (ойтика), Lupinus angustifolius (люпин), вид Miscanthus, такой как Miscanthus x giganteus и Miscanthus sinensis, Panicum virgatum (просо прутьевидное), Pongamia pinnata (индийский бук), Populus trichocarpa, Ricinus communis (клещевина), вид Saccharum (сахарный тростник), Sesamum indicum (кунжут), Solanum tuberosum (картофель), вид Sorghum такой как Sorghum bicolor, Sorghum vulgare, Theobroma grandiforum (какао) вид Trifolium, и вид Triticum. (пшеница) такой как Triticum aestivum. Растительное масло может быть экстрагировано из вегетативных частей растения любым способом, известным из уровня техники. Такие способы обычно включают экстракцию неполярными растворителями, такими как диэтиловый эфир, петролейный эфир, смеси хлороформ/метанол или бутанол, в общем, ассоциирующимися с первым дроблением семян. Липиды, ассоциированные с крахмалом или другими полисахаридами, могут быть экстрагированы с помощью насыщенного водой бутанола. Масло из семян может быть «дегуммировано» способами, известными в данной области, чтобы удалить полярные липиды, такие как фософолипиды, или обработано другими способами, чтобы удалить загрязняющие вещества или улучшить чистоту, стабильность или цвет. TAG и другие сложные эфиры в растительном масле могут быть гидролизованы с целью высвобождения свободных жирных кислот, или масло может быть гидрогенизировано, обработано химическим или ферментным способом, как известно в данной области. Как используют в настоящем описании, термин «масло из семян» имеет аналогичное значение, за исключением того, что он относится к композиции липидов, полученной из семян растений по изобретению.
В настоящем описании термин «жирная кислота» обозначает карбоновую кислоту с алифатическим хвостом размером по меньшей мере 8 атомов углерода, насыщенным или ненасыщенным. Предпочтительные жирные кислоты содержат углеродно-углеродную цепь длиной по меньшей мере 12 атомов углерода. Большинство природных жирных кислот содержат четное количество атомов углерода, поскольку в их биосинтезе участвует ацетат, содержащий два атома углерода. Жирные кислоты могут находиться в свободном состоянии (неэтерифицированные) или в этерифицированной форме, например, как часть TAG, DAG, MAG, связи ацил-CoA (тиоэфир), связи ацил-ацилпереносящий белок (ACP) или другой ковалентно связанной форме. Являясь ковалентно связанной в этерифицированной форме, жирная кислота в настоящем описании обозначена как группа «ацил». Жирная кислота может быть этерифицирована как фосфолипид, например, фосфатидилхолин (PC), фосфатидилэтаноламин (PE), фосфатидилсерин (PS), фосфатидилглицерил (PG), фосфатидилинозитол (PI) или дифосфатидилглицерил. Насыщенные жирные кислоты не содержат двойных связей или других функциональных групп на протяжении цепи. Термин «насыщенный» относится к водороду, в том смысле, что все атомы углерода (кроме карбоксильной группы [-COOH]) связаны с максимально возможным количеством атомов водорода. Иными словами, омега (ω) конец содержит 3 атома водорода (CH3-), и каждый атом углерода в цепи связан с 2 атомами водорода (-CH2-). Ненасыщенные жирные кислоты по форме сходны с насыщенными жирными кислотами, за исключением того, что одна или несколько алкеновых функциональных групп присутствуют на протяжении цепи, где каждый алкен замещает часть одинарной связи «-CH2-CH2-» в цепи частью двойной связи «-CH=CH-» (т. е., атом углерода связан двойной связью с другим атомом углерода). Два следующих атома углерода в цепи, присоединенные с каждой стороны от двойной связи, могут находиться в цис- или транс-конфигурации.
В настоящем описании термин «мононенасыщенная жирная кислота» или «MUFA» обозначает жирную кислоту, содержащую по меньшей мере 12 атомов углерода в углеродной цепи и только одну алкеновую группу (двойная углерод-углеродная связь), которая может находиться в этерифицированной или неэтерифицированной (свободной) форме. В настоящем описании термин «полиненасыщенная жирная кислота» или «PUFA» обозначает жирную кислоту, содержащую по меньшей мере 12 атомов углерода в углеродной цепи и по меньшей мере две алкеновые группы (двойные углерод-углеродные связи), которая может находиться в этерифицированной или неэтерифицированной форме.
«Моноацилглицерид» или «MAG» представляет собой глицерид, в котором глицерин этерифицирован одной жирной кислотой. В настоящем описании MAG содержит гидроксильную группу в положении sn-1/3 (также обозначено в настоящем описании как sn-1 MAG или 1-MAG, или 1/3-MAG) или sn-2 (также обозначено в настоящем описании как 2-MAG), и, таким образом, MAG не содержит фосфорилированных молекул, таких как PA или PC. MAG, таким образом, является компонентом нейтральных липидов в растении или его части.
«Диацилглицерид» или «DAG» представляет собой глицерид, в котором глицерин этерифицирован двумя жирными кислотами, которые могут быть одинаковыми или, предпочтительно, разными. В настоящем описании DAG содержит гидроксильную группу в положении sn-1,3 или sn-2, и, таким образом, DAG не содержит фосфорилированных молекул, таких как PA или PC. DAG, таким образом, является компонентом нейтральных липидов в растении или его части. В пути Кеннеди синтеза DAG (Фигура 1), предшественник sn-глицерил-3-фосфат (G-3-P) этерифицируется двумя ацильными группами, каждая из которых поступает от сложного эфира жирной кислоты кофермента A, при этом, первая реакция катализируется глицерин-3-фосфат-ацилтрансферазой (GPAT) в положении sn-1 с образованием LysoPA, с последующей реакцией присоединения второго ацила в положении sn-2, катализированного ацилтрансферазой лизофосфатидиновой кислоты (LPAAT), с образованием фосфатидиновой кислоты (PA). Данное промежуточное соединение затем дефосфорилируется PAP с образованием DAG. Кроме того, DAG может быть образован из TAG удалением ацильной группы под действием липазы или из PC по существу удалением головной группы холина посредством любого из ферментов PDCT, PLC или PLD (Фигура 1).
«Триацилглицерид» или «TAG» представляет собой глицерид, в котором глицерин этерифицирован тремя жирными кислотами, которые могут быть одинаковыми (например, как в триолеине) или, чаще, разными. В пути Кеннеди синтеза TAG, DAG образуется, как описано выше, а затем третья ацильная группа присоединяется к остову глицерина под действием DGAT. Альтернативные пути образования TAG включают путь, катализированный ферментом PDAT (Фигура 1) и путь MGAT, описанные в настоящем описании.
В настоящем описании термин «дикий тип» или его варианты обозначают клетку, растение или его часть, такую как клетка, вегетативная часть растения, семя, клубень или свекла, которые не были генетически модифицированы, например, клетки, растения или их части, не содержащие первого и второго экзогенных полинуклеотидов по настоящему изобретению.
Термин «соответствующий» обозначает клетку, растение или его часть, такую как клетка, вегетативная часть растения, семя, клубень или свекла, которые имеют одинаковый или сходный генетический фон с клеткой, растением или его частью, такой как вегетативная часть растения, семя, клубень или свекла по изобретению, но при этом они не были модифицированы, как описано в настоящем описании (например, вегетативная часть растения или семя, лишенные первого и второго экзогенных полинуклеотидов). В предпочтительном варианте осуществления, соответствующие растение или его часть, такая как вегетативная часть растения, находятся на той же стадии развития, что и растение или его часть, такая как вегетативная часть растения, по изобретению. Например, если растение представляет собой цветущее растение, то соответствующее растение также, предпочтительно, находится в стадии цветения. Соответствующие клетку, растение или его часть, такую как вегетативная часть растения, можно использовать в качестве контроля для сравнения уровней нуклеиновой кислоты или экспрессии белка, или степени и природы модификации признака, например, TTQ и/или содержания TAG, с клеткой, растением или его частью, такой как вегетативная часть растения по изобретению, модифицированными, как описано в настоящем описании. Специалист в данной области может легко определять подходящие «соответствующие» клетку, растение или его часть, такую как вегетативная часть растения, для такого сравнения.
Как используют в настоящем описании, «по сравнению с» или «относительно» обозначает сравнение уровней, например, TTQ или содержания триацилглицерина (TAG), одного или нескольких, или всех из содержания растворимого белка, содержания азота, соотношения углерод:азот, экспрессии фотосинтетического гена, способности к фотосинтезу, общего содержания пищевых волокон (TDF), содержания углерода и содержания энергии, или содержания или состава неполярного липида, общего содержания неполярных липидов, общего содержания жирных кислот или другого параметра клетки, растения или его части, содержащих один или несколько экзогенных полинуклеотидов, генетических модификаций или экзогенных полипептидов, с клеткой, растением или его частью, такой как вегетативная часть растения, лишенных одного или нескольких экзогенных полинуклеотидов, генетических модификаций или полипептидов.
В настоящем описании каждый из «синергизма», «синергического», «действующих синергически» и родственных терминов представляет собой сравнительный термин, который означает, что эффект комбинации элементов, присутствующих в растении или его части по изобретению, например, комбинации элементов A и B, превышает сумму эффектов элементов по отдельности в соответствующих растениях или их частях, например, сумму эффекта A и эффекта B. Когда более двух элементов присутствуют в растении или его части, например, элементы A, B и C, это означает, что эффект комбинации всех элементов превышает сумму эффектов индивидуальных элементов. В предпочтительном варианте осуществления это означает, что эффект комбинации элементов A, B и C превышает сумму объединенного эффекта элементов A и B и эффекта элемента C. В таком случае, может быть указано, что элемент C действует синергически с элементами A и B. Необходимо понимать, что эффекты измеряют в соответствующих клетках, растениях или их частях, например, выращенных в таких же условиях и находящихся на такой же стадии биологического развития.
В настоящем описании, «прорастать со скоростью, в основном такой же, как и соответствующее растение дикого типа», или подобные фразы, означают, что семя растения по изобретению относительно способно к прорастанию при сравнении с семенем растения дикого типа, не содержащего определенных экзогенного полинуклеотида(полинуклеотидов) и генетических модификаций. Прорастание можно измерять in vitro на питательной среде для культуры ткани или в грунте, как это происходит в поле. В одном варианте осуществления количество семян, которые прорастают, например, при выращивании в оптимальных условиях теплицы для данного вида растений, составляет по меньшей мере 75%, более предпочтительно, по меньшей мере 90%, по сравнению с соответствующими семенами дикого типа. В другом варианте осуществления семена, которые прорастают, например, при выращивании в оптимальных условиях теплицы для данного вида растений, дают саженцы, растущие со скоростью, которая, в среднем, составляет по меньшей мере 75%, более предпочтительно, по меньшей мере 90%, по сравнению с соответствующими растениями дикого типа. Это обозначено как «сила роста саженцев». В одном варианте осуществления, скорость начального роста корней и роста побегов у саженцев по изобретению является по существу такой же, как у соответствующего саженца дикого типа, выращенного в таких же условиях. В одном варианте осуществления, биомасса листьев (сухая масса) растений по изобретению составляет по меньшей мере 80%, предпочтительно, по меньшей мере 90% относительно биомассы листьев соответствующих растений дикого типа, выращенных в таких же условиях, предпочтительно, в поле. В одном варианте осуществления, высота растений по изобретению составляет по меньшей мере 70%, предпочтительно, по меньшей мере 80%, более предпочтительно, по меньшей мере 90% высоты растения относительно соответствующего растения дикого типа, выращенного в таких же условиях, предпочтительно, в поле и предпочтительно, в состоянии зрелости.
Как используют в настоящем описании, термин «экзогенный полинуклеотид, осуществляющий понижающую регуляцию продукции и/или активности эндогенного полипептида», или его варианты, относится к полинуклеотиду, который кодирует молекулу РНК, обозначенную в настоящем описании как «осуществляющая сайленсинг молекула РНК», или ее варианты (например, кодирующему имиРНК или шпРНКи), осуществляющие понижающую регуляцию продукции и/или активности, или самостоятельно осуществляет понижающую регуляцию продукции и/или активности (например, представляет собой имиРНК или шпРНК, которые можно доставлять непосредственно, например, в растение или его часть) эндогенного полипептида. Это включает случай, когда исходный транскрипт РНК, полученный посредством экспрессии экзогенного полинуклеотида, подвергается процессингу в клетке с образованием фактически осуществляющей сайленсинг молекулы РНК. эндогенные полипептиды, продукция или активность которых подвергается понижающей регуляции, включают, например, TAG-липазу SDP1, пластидную GPAT, пластидную LPAAT, полипептид TGD, такой как TGD5, TST, такой как TST1 или TST2, АГФазу, PDCT, CPT или десатуразу жирных кислот Δ12 (FAD2), или комбинацию двух или более из них. Как правило, молекула РНК уменьшает экспрессию эндогенного гена, кодирующего полипептид. Степень понижающей регуляции составляет, как правило, менее чем 100%, например, продукция или активность уменьшена на между 25% и 95% по сравнению с диким типом. Оптимальный уровень остаточной продукции или активности можно определять общепринятым способом.
В настоящем описании термин «в пересчете на массу» обозначает массу вещества (например, TAG, DAG, жирной кислоты, белка, азота, углерода) как процент от массы композиции, содержащей вещество (например, сухой массы семени, листа). Например, если трансгенное семя содержит 25 мкг общих жирных кислот на 120 мкг массы семени, процент от общих жирных кислот в пересчете на массу составляет 20,8%.
В настоящем описании термин «в относительном выражении» обозначает параметр, например, количество вещества в композиции, содержащей вещество, в сравнении с параметром для соответствующей композиции, в процентном выражении. Например, уменьшение с 3 единиц до 2 единиц является уменьшением на 33% в относительном выражении.
В настоящем описании «пластиды» представляют собой органеллы в растениях, включая морские водоросли, которые являются местом продукции углеродных соединений на основе фотосинтеза, включая сахара, крахмал и жирные кислоты. Пластиды включают хлоропласты, содержащие хлорофилл и осуществляющие фотосинтез, этиопласты, которые являются предшественниками хлоропластов, а также специализированные пластиды, такие как хромопласты, которые представляют собой окрашенные пластиды для синтеза и хранения пигментов, геронтопласты, которые руководят демонтажем фотосинтетических структур в ходе старения, амилопласты для синтеза и хранения крахмала, элайопласты для хранения липидов и протеинопласты для хранения и модификации белков.
В настоящем описании термин «биологическое топливо» обозначает любой вид топлива, обычно применяемый для продукции энергии машинным оборудованием, например, автомобилями, самолетами, катерами, большими грузовыми машинами или работающими на нефти двигателями, энергия которого происходит от биологической фиксации углерода. Биотопливо включает виды топлива, получаемые в результате превращения биомассы, а также твердой биомассы, жидких видов топлива и биогазов. Примеры биологического топлива включают биоспирты, биодизельное топливо, синтетическое дизельное топливо, вегетативное масло, биоэфиры, биогаз, синтетический газ, твердое биологическое топливо, полученное из водорослей топливо, биоводород, биометанол, 2,5-диметилфуран (DMF), биодиметиловый эфир (биоDME), дизельное топливо Фишера-Тропша, биоводородное дизельное топливо, смешанные спирты и древесное дизельное топливо.
В настоящем описании термин «биоспирт» обозначает полученные биологическим способом спирты, например, этанол, пропанол и бутанол. Биоспирты образуются под действием микроорганизмов и/или ферментов посредством ферментации сахаров, гемицеллюлозы или целлюлозы.
В настоящем описании термин «биодизельное топливо» обозначает состав, содержащий метиловые или этиловые эфиры жирной кислоты, полученные из липидов переэтерификацией, где липиды получены из живых клеток, а не из окаменелых остатков.
В настоящем описании термин «синтетическое дизельное топливо» обозначает форму дизельного топлива, которое получено из возобновляемого сырья вместо окаменелого сырья, используемого в большинстве видов дизельного топлива.
В настоящем описании термин «растительное масло» включает чистое растительное масло (или масло прямого отжима) или масло из отходов (побочный продукт других отраслей промышленности), включая масло, которое продуцируется в вегетативной части или семени растения. Растительное масло включает масло из вегетативных частей растений или масло из семян, как определено в настоящем описании.
В настоящем описании термин «биогаз» обозначает метан или легковоспламеняющуюся смесь метана и других газов, полученные анаэробным расщеплением органического материала анаэробами.
В настоящем описании термин «синтетический газ» обозначает горючую смесь, которая содержит меняющиеся количества монооксида углерода и водорода, и может содержать другие углеводороды, полученные частичным окислением биомассы. Синтетический газ можно переводить в метанол в присутствии катализатора (обычно на основе меди), с последующей дегидратацией метанола в присутствии другого катализатора (например, диоксида кремния-алюминия).
В настоящем описании термин «биоуголь» обозначает древесный уголь, полученный из биомассы, например, пиролизом биомассы.
В настоящем описании термин «сырье» обозначает материал, например, биомассу или продукт ее превращения (например, синтетический газ), который используют для производства продукта, например, биологического топлива, например, биодизельного топлива или синтетического дизельного топлива.
В настоящем описании термин «промышленный продукт» обозначает углеводородный продукт, который в основном состоит из атомов углерода и водорода, например, такой как метиловые и/или этиловые эфиры жирной кислоты, или алканы, такие как метан, смеси алканов с более длинной цепью, которые обычно являются жидкостями при температуре окружающей среды, биологическое топливо, монооксид углерода и/или водорода или биоспирт, например, этанол, пропанол или бутанол или биоуголь. Термин «промышленный продукт» предназначен включать промежуточные продукты, которые могут быть превращены в другие промышленные продукты, например, синтетический газ, который сам по себе рассматривается, как промышленный продукт, может использоваться для синтеза углеводородного продукта, который также рассматривается, как промышленный продукт. Термин «промышленный продукт» в настоящем описании включает обе чистые формы упомянутых выше соединений или, чаще, смесь различных соединений и компонентов, например, углеводородный продукт может содержать соединения с длиной углеродной цепи в определенном интервале, как хорошо известно в данной области.
В настоящем описании «потомок» обозначает ближайшее и все последующие поколения потомства, полученные от родительского организма, например, второе, третье или более поздние поколения потомства.
Как используют в настоящем описании, термин «предок» относится к любому более раннему поколению растения, содержащему первый и второй экзогенные полинуклеотиды. Предок может представлять собой родительское растение, прародительское растение, прапрародительское растение и т.д.
Как используют в настоящем описании, термин «отбор растения» обозначает активный отбор растения на основании того, что оно имеет желательный фенотип, такой как увеличенное TTQ, увеличенное содержание TAG и белка, по сравнению с соответствующим растением дикого типа.
Как используют в настоящем описании, такие фразы, как «имеют содержание TFA приблизительно 5% (масс./масс. сухой массы)», или «имеют общее содержание TAG приблизительно 6% (масс./масс. сухой массы)», или сходным образом структурированные фразы, означают, что может присутствовать больше определенного уровня. Например, фразу «имеют содержание TFA приблизительно 5% (масс./масс. сухой массы)» можно использовать взаимозаменяемо с «содержит по меньшей мере приблизительно 5% TFA (масс./масс. сухой массы)». Расширяя этот пример далее, вегетативная часть растения, которая имеет содержание TFA приблизительно 5% (масс./масс. сухой массы), может иметь 6% или 7,5%, или более высокое содержание TFA.
Как используют в настоящем описании, если контекст не требует иного, термин «увеличенное содержание», при использовании применительно к полипептиду, или сходные фразы, включающие ссылку на конкретный полипептид, относится либо к экзогенному полипептиду, либо к эндогенному полипептиду. Например, вегетативная часть растения по изобретению может иметь увеличенное содержание полипептида WRI1, увеличенное содержание полипептида DGAT и уменьшенное содержание полипептида SDP1, каждое по сравнению с соответствующей вегетативной частью растения дикого типа, где каждый из полипептидов WRI1 и DGAT независимо представляет собой либо экзогенный полипептид, либо эндогенный полипептид. В качестве другого примера, вегетативная часть растения по изобретению может иметь увеличенное содержание полипептида WRI1, увеличенное содержание полипептида DGAT и увеличенное содержание полипептида LEC2, каждое по сравнению с соответствующей вегетативной частью растения дикого типа, где каждый из полипептидов WRI1, DGAT и LEC2 независимо представляет собой либо экзогенный полипептид, либо эндогенный полипептид. В качестве следующего примера, a вегетативная часть растения по изобретению может иметь увеличенное содержание полипептида PDAT или DGAT, уменьшенное содержание полипептида TGD и уменьшенное содержание полипептида SDP1, каждое по сравнению с соответствующей вегетативной частью растения дикого типа где PDAT или DGAT представляет собой либо экзогенный полипептид, либо эндогенный полипептид и так далее. Экзогенный полипептид может являться результатом экспрессии трансгена, кодирующего полипептид в клетке или растении, или его части по изобретению. Эндогенный полипептид может являться результатом увеличенной экспрессии эндогенного гена, например, индукции сверхэкспрессии и/или обеспечения увеличенных уровней фактора(факторов) транскрипции для гена.
В настоящем описании слово «содержат» или его варианты, например, «содержит» или «содержащий» следует понимать, как обозначающее включение указанного элемента, целого числа или стадии, или группы элементов, целых чисел или стадий, но не исключение любого другого элемента, целого числа или стадии, или группы элементов, целых чисел или стадий.
Термин «и/или», например, «X и/или Y» следует понимать, как обозначающий любое из «X и Y» или «X или Y» и интерпретировать как обеспечивающий явное присутствие обоих значений, или любого из значений.
В настоящем описании термин приблизительно, если не указано иное, обозначает +/- 10%, более предпочтительно, +/- 5%, более предпочтительно, +/- 2%, более предпочтительно, +/- 1%, даже более предпочтительно, +/- 0,5% от указанного значения.
Получение растений с модифицированными признаками
Настоящее изобретение основано на открытии того факта, что признаки растений, такие как два или более из содержания неполярных липидов, содержания белка, TTQ, содержания TAG, содержания азота, содержания углерода, в растениях или их частях можно улучшать с помощью комбинации модификаций, выбранных из обозначенных в настоящем описании как: (A). Push, (B). Pull, (C). Protect, (D). Package, (E). Plastidial export, (F). Plastidial import и (G). Prokariotyc Pathway.
Растения или их части, такие как вегетативные части растений, по изобретению, таким образом, имеют ряд комбинаций экзогенных полинуклеотиды и/или генетических модификаций, каждая из которых обеспечивает одну из модификаций. Эти экзогенные полинуклеотиды и/или генетические модификации включают:
(A) экзогенный полинуклеотид, кодирующий полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в растении или его части, такой как вегетативная часть растения, обеспечивающий модификацию «Push»,
(B) экзогенный полинуклеотид, кодирующий полипептид, вовлеченный в биосинтез одного или нескольких неполярных липидов в растении или его части, такой как вегетативная часть растения, обеспечивающий модификацию «Pull»,
(C) генетическую модификацию, осуществляющую понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в катаболизм триацилглицеринов (TAG) в растении или его части, такой как вегетативная часть растения, по сравнению с соответствующим растением или его частью, такой как вегетативная часть растения, лишенными генетической модификации, обеспечивающую модификацию «Protect»,
(D) экзогенный полинуклеотид, кодирующий полипептид покрытия масляных телец (OBC), такой как ассоциированный с липидными капельками полипептид (LDAP), обеспечивающий модификацию «Package»,
(E) экзогенный полинуклеотид, кодирующий полипептид, который увеличивает экспорт жирных кислот из пластид в растении или его части, такой как вегетативная часть растения, по сравнению с соответствующим растением или его частью, такой как вегетативная часть растения, лишенными экзогенного полинуклеотида, обеспечивающий модификацию «Plastidial export»,
(F) генетическую модификацию, осуществляющую понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в импорт жирных кислот в пластиды в растении или его части, такой как вегетативная часть растения, по сравнению с соответствующим растением или его частью, такой как вегетативная часть растения, лишенными генетической модификации, обеспечивающую модификацию «Plastidial import», и
G) генетическую модификацию, осуществляющую понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в продукцию диацилглицерина (DAG) в пластиде в растении или его части, такой как вегетативная часть растения, по сравнению с соответствующим растением или его частью, такой как вегетативная часть растения, лишенными генетической модификации, обеспечивающую модификацию «Prokaryotic pathway».
Предпочтительные комбинации (дополнительно обозначенные в настоящем описании как наборы) экзогенных полинуклеотидов и/или генетических модификаций по изобретению представляют собой;
1) A, B и необязательно, одно из C, D, E, F или G;
2) A, C и необязательно, одно из D, E, F или G;
3) A, D и необязательно, одно из E, F или G;
4) A, E и необязательно F или G;
5) A, F и необязательно G;
6) A и G;
7) A, B, C и необязательно, одно из D, E, F или G;
8) A, B, D и необязательно, одно из E, F или G;
9) A, B, E и необязательно F или G;
10) A, B, F и необязательно G;
11) A, B, C, D и необязательно, одно из E, F или G;
12) A, B, C, E и необязательно, F или G;
13) A, B, C, F и необязательно, G;
14) A, B, D, E и необязательно, F или G;
15) A, B, D, F и необязательно, G;
16) A, B, E, F и необязательно, G;
17) A, C, D и необязательно, одно из E, F или G;
18) A, C, E и необязательно, F или G;
19) A, C, F и необязательно, G;
20) A, C, D, E и необязательно, F или G;
21) A, C, D, F и необязательно, G;
22) A, C, E, F и необязательно, пятая модификация G;
23) A, D, E и необязательно, F или G;
24) A, D, F и необязательно, G;
25) A, D, E, F и необязательно, G;
26) A, E, F и необязательно G;
27) шесть из A, B, C, D, E, F и G, за исключением одного из A, B, C, D, E, F или G, и
28) любая из 1-26 выше, где присутствуют два или более экзогенных полинуклеотида, кодирующие два или более различных полипептида факторов транскрипции, которые увеличивают экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в растении или его части, например, один экзогенный полинуклеотид, кодирующий WRI1 и другой экзогенный полинуклеотид, кодирующий LEC2.
В каждой из приведенных выше предпочтительных комбинаций могут присутствовать по меньшей мере два различных экзогенных полинуклеотида, кодирующие по меньшей мере два различных полипептида факторов транскрипции, которые увеличивают экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в растении или его части, такой как вегетативная часть растения.
Эти модификации более полно описаны, как указано ниже:
A. Модификация «Push (Толчок)» отличается повышенным синтезом общих жирных кислот в пластидах в растении или его части. В одном варианте осуществления, это происходит благодаря повышенной экспрессии и/или активности фактора транскрипции, который регулирует синтез жирных кислот в пластидах. В одном варианте осуществления, это может быть достигнуто посредством экспрессии в трансгенном растении или его части экзогенного полинуклеотида, кодирующий полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в растении или его части. В одном варианте осуществления, повышенный синтез жирных кислот не является вызванным доставкой в растение или его часть модифицированной ACCазы, активность которой в меньшей степени ингибируется жирными кислотами, по сравнению с эндогенной ACCазой в растении или его части. В одном варианте осуществления, растение или его часть содержит экзогенный полинуклеотид, кодирующий фактор транскрипции, предпочтительно, под контролем промотора, отличного от конститутивного промотора. Фактор транскрипции может быть выбран из группы, состоящей из WRI1, LEC1, LEC1-подобного, LEC2, BBM, FUS3, ABI3, ABI4, ABI5, Dof4, Dof11 или из группы, состоящей из MYB73, bZIP53, AGL15, MYB115, MYB118, TANMEI, WUS, GFR2a1, GFR2a2 и PHR1, и предпочтительно, представляет собой WRI1, LEC1 или LEC2. В следующем варианте осуществления, увеличенный синтез общих жирных кислот присутствует по сравнению с соответствующим растением дикого типа или его частью. В одном варианте осуществления, присутствует два или более экзогенных полинуклеотида, кодирующих два или более различных полипептидов факторов транскрипции. Модификация «Push» может также быть достигнута посредством увеличенной экспрессии полипептидов, модулирующих активность WRI1, таких как полипептиды MED15 или 14-3-3.
B. Модификация «Pull (Притягивание)» отличается повышенной экспрессией и/или активностью в растении или его части ацилтрансферазы жирных ацилов, катализирующей синтез TAG, DAG или MAG в растении или его части, такой как DGAT, PDAT, LPAAT, GPAT или MGAT, предпочтительно, DGAT или PDAT. В одном варианте осуществления, это может быть достигнуто посредством экспрессии в трансгенном растении или его части экзогенный полинуклеотид, кодирующий полипептид, вовлеченный в биосинтез одного или нескольких неполярных липидов. В одном варианте осуществления, ацилтрансфераза представляет собой связанную с мембраной ацилтрансферазу, которая использует субстрат ацил-CoA в качестве донора ацила в случае в случае DGAT, LPAAT, GPAT или MGAT, или ацильную группу из PC в качестве донора ацила в случае PDAT. Модификация Pull может присутствовать по сравнению с соответствующим растением дикого типа или его частью или, предпочтительно, по сравнению с соответствующим растением или его частью, содержащими модификацию Push. В одном варианте осуществления, растение или его часть содержит экзогенный полинуклеотид, кодирующий ацилтрансферазу жирных ацилов. Модификация «Pull» может также быть достигнута посредством увеличенной экспрессии полипептида PDCT, CPT или фосфолипазы C или D, которая увеличивает продукцию DAG из PC.
C. Модификация «Protect (Защита)» отличается снижением катаболизма триацилглицеринов (TAG) в растении или его части. В одном варианте осуществления, это может быть достигнуто посредством генетической модификации в растении или его части, осуществляющую понижающую регуляцию продукции и/или активности полипептида, вовлеченного в катаболизм триацилглицеринов (TAG) в растении или его части, по сравнению с соответствующими растением или его частью, лишенными генетической модификации. В одном варианте осуществления, растение или его часть имеет сниженную экспрессию и/или активность эндогенной TAG-липазы в растении или его части, предпочтительно, липазы SDP1, полипептида Cgi58, ацил-CoA-оксидазы, такой как ACX1 или ACX2, или полипептида, вовлеченного в β-окисление жирных кислот в растении или его части, такого как пероксисомальный транспортер АТФ-связывающей кассеты PXA1. Это может происходить благодаря экспрессии в растении или его части экзогенного полинуклеотида, кодирующего молекулу РНК, которая уменьшает экспрессию, например, эндогенного гена, кодирующего TAG-липазу, такую как липаза SDP1, ацил-CoA-оксидазу или полипептид, вовлеченный в β-окисление жирных кислот в растении или его части, или посредством мутации в эндогенном гене, кодирующем, например, TAG-липазу, ацил-CoA-оксидазу или полипептид, вовлеченный в β-окисление жирных кислот. В одном варианте осуществления, сниженная экспрессия и/или активность присутствует по сравнению с соответствующим растением дикого типа или его частью или по сравнению с соответствующим растением или его частью, имеющими модификацию Push.
D. Модификация «Package (Упаковка)» отличается увеличенными экспрессией и/или накоплением полипептида покрытия масляных телец (OBC). В одном варианте осуществления, это может быть достигнуто посредством экспрессии в трансгенном растении или его части экзогенного полинуклеотида, кодирующего полипептид покрытия масляных телец (OBC). Полипептид OBC может представлять собой олеозин, например, такой как полиолеозин, каолеозин или стеролеозин, или предпочтительно, LDAP. В одном варианте осуществления, уровень олеозина, который накапливается в растении или его части, по меньшей мере в 2 раза выше по сравнению с соответствующим растением или его частью, содержащими ген олеозина из T-ДНК pJP3502. В одном варианте осуществления, увеличенные экспрессия или накопление полипептида OBC не являются вызванными исключительно модификацией Push. В одном варианте осуществления, экспрессия и/или накопление присутствует по сравнению с соответствующим растением дикого типа или его частью или, предпочтительно, по сравнению с соответствующим растением или его частью, имеющими модификацию Push.
E. Модификация «Plastidial export (Пластидный экспорт)» отличается повышенной скоростью экспорта общих жирных кислот из пластид в растении или его части. В одном варианте осуществления, это может быть достигнуто посредством экспрессии в растении или его части экзогенного полинуклеотида, кодирующего полипептид, который увеличивает экспорт жирных кислот из пластид в растении или его части по сравнению с соответствующим растением или его частью, лишенным экзогенного полинуклеотида. В одном варианте осуществления, это происходит благодаря увеличенной экспрессии и/или активности тиоэстеразы (TE) жирных кислот, полипептида транспортера жирных кислот, такого как полипептид ABCA9, или длинноцепочечной ацил-CoA-синтетазы (LACS). В одном варианте осуществления, растение или его часть содержит экзогенный полинуклеотид, кодирующий TE, полипептид транспортера жирных кислот или LACS. TE может представлять собой полипептид FATB или, предпочтительно, полипептид FATA. В одном варианте осуществления, модификация Plastidial export присутствует по сравнению с соответствующим растением дикого типа или его частью или, предпочтительно, по сравнению с соответствующим растением или его частью, имеющими модификацию Push.
F. Модификация «Plastidial import (Пластидный импорт)» отличается сниженной скоростью импорта жирных кислот в пластиды в растении или его части из пространства за пределами пластид. В одном варианте осуществления, это может быть достигнуто с помощью генетической модификации в растении или его части, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в импорт жирных кислот в пластиды в растении или его части, по сравнению с соответствующими растением или его частью, лишенными генетической модификации. Например, это может происходить благодаря экспрессии в растении или его части экзогенного полинуклеотида, кодирующего молекулу РНК, которая уменьшает экспрессию эндогенного гена, кодирующего полипептид транспортера, такой как полипептид TGD, например, a TGD1, TGD2, TGD3, TGD4 или предпочтительно, полипептид TGD5, или посредством мутации в эндогенном гене, кодирующем полипептид TGD. В одном варианте осуществления, уменьшенная скорость импорта присутствует по сравнению с соответствующим растением дикого типа или его частью или по сравнению с соответствующим растением или его частью, имеющими модификацию Push.
G. Модификация «Prokaryotic pathway (Прокариотический путь)» отличается уменьшенным количеством DAG или уменьшенной скоростью продукции DAG в пластидах в растении или его части. В одном варианте осуществления, это может быть достигнуто посредством генетической модификации в растении или его части, осуществляющей понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в продукцию диацилглицерина (DAG) в пластиде, по сравнению с соответствующими растением или его частью, лишенными генетической модификации. В одном варианте осуществления, уменьшенные количество или скорость продукции DAG является результатом сниженной продукции LPA из ацил-ACP и G3P в пластидах. Сниженные количество или скорость продукции DAG может быть результатом экспрессии в растении или его части экзогенного полинуклеотида, кодирующего молекулу РНК, которая уменьшает экспрессию эндогенного гена, кодирующего пластидную GPAT, пластидную LPAAT или пластидную PAP, предпочтительно, пластидную GPAT, или посредством мутации в эндогенном гене, кодирующем пластидный полипептид. В одном варианте осуществления, уменьшенные количество или скорость продукции DAG присутствуют по сравнению с соответствующим растением дикого типа или его частью или, предпочтительно, по сравнению с соответствующим растением или его частью, имеющими модификацию Push.
Модификация Push является очень желательной по изобретению, а модификация Pull является предпочтительной. Модификации Protect и Package могут быть комплементарными, т.е. одной из двух может быть достаточно. Растение или его часть может содержать одну, две или все три из модификаций Plastidial export, Plastidial import и Prokaryotic pathway. В одном варианте осуществления, по меньшей мере один из экзогенных полинуклеотидов в растении или его части, предпочтительно, по меньшей мере экзогенный полинуклеотид, кодирующий фактор транскрипции, который регулирует синтез жирных кислот в пластидах, экспрессируется под контролем (H) промотора, отличного от конститутивного промотора, например, такого как связанный с развитием промотор, промотор, который предпочтительно, активен в фотосинтезирующих клетках, тканеспецифический промотор, промотор, модифицированный посредством снижения уровня его экспрессии по сравнению с соответствующим нативным промотором, или предпочтительно, промотора, представляющего собой специфический для старения промотор. Более предпочтительно, по меньшей мере экзогенный полинуклеотид, кодирующий фактор транскрипции, который регулирует синтез жирных кислот в пластидах, экспрессируется под контролем промотора, отличного от конститутивного промотора, и экзогенный полинуклеотид, кодирующий молекулу РНК, осуществляющую понижающую регуляцию эндогенной продукции и/или активности полипептида, вовлеченного в катаболизм триацилглицеринов, также экспрессируется под контролем промотора, отличного от конститутивного промотора, где промоторы могут быть одинаковыми или различными. Альтернативно, в однодольных растениях, экзогенный полинуклеотид, кодирующий фактор транскрипции, который регулирует синтез жирных кислот в пластидах, экспрессируется под контролем конститутивного промотора, например, такой как промотор гена убиквитина или промотор гена актина.
Растения продуцируют некоторые, но не все из их мембранных липидов, такие как MGDG, в пластидах по так называемому прокариотическому пути (Фигура 1). Кроме того, в растениях присутствует эукариотический путь для синтеза галактолипидов и глицеролипидов, по которому FA сначала синтезируются в пластиде, а затем в ER происходит сборка FA в глицеролипиды. MGDG, синтезированный по эукариотическому пути, содержит C18:3 жирную кислоту (ALA), этерифицированную в положении sn-2 MGDG. Сборка остова DAG, содержащего ALA, для синтеза MGDG по этому пути, происходит в ER, после чего происходит его импорт в пластиду. В противоположность этому, MGDG, синтезированный по прокариотическому пути, содержит C16:3 жирную кислоту, этерифицированную в положении sn-2 MGDG. Соотношение вклада прокариотического пути относительно эукариотического пути в продукцию MGDG (16:3) против MGDG (18:3) представляет собой характерный и отличительный признак различных видов растений (Mongrand et al. 1998). Этот отличительный состав жирных кислот MGDG позволяет классифицировать все высшие растения (покрытосеменные) как так называемые 16:3 или 18:3 растения. Виды 16:3, примером которых являются Arabidopsis и Brassica napus, как правило, имеют активные прокариотический и активный эукариотический пути синтеза MGDG, тогда как виды 18:3, примером которых являются Sorghum bicolor, Zea mays, Nicotiana tabacum, Pisum sativum и Glycine max, как правило, имеют только (или почти полностью) эукариотический путь синтеза MGDG, с небольшим или отсутствующей накоплением C16:3 жирной кислоты в вегетативных тканях. Как используют в настоящем описании, «растение 16:3» или «вид 16:3» представляет собой такое, которое содержит более чем 2% C16:3 жирной кислоты в общем содержании жирных кислот в его фотосинтезирующих тканях. Как используют в настоящем описании, «растение 18:3» или «вид 18:3» представляет собой такое, которое содержит менее чем 2% C16:3 жирных кислот в общем содержании жирных кислот в его фотосинтезирующих тканях. Как описано в настоящем описании, растение можно переводить из растения 16:3 в растение 18:3 посредством подходящих генетических модификаций. Соотношение потока между прокариотическим и эукариотическим путями не является консервативным для разных видов растений или тканей. В видах 16:3 до 40% потока в листьях проходит по прокариотическому пути (Browse et al., 1986), тогда как в видах 18:3, таких как горох и соя, приблизительно 90% FA, которые синтезированы в пластиде, экспортируется из пластиды в ER, чтобы обеспечить источник FA для эукариотического пути (Ohlrogge and Browse, 1995; Somerville et al., 2000).
Таким образом, различные количества 18:3 и 16:3 жирных кислот обнаружены в гликолипидах различных видов растений. Это используют для того, чтобы отличать растения 18:3, в которых жирные кислоты с 3 двойными связями практически полностью являются C18 жирными кислотами, и растения 16:3, которые содержат как C16-, так и C18-жирные кислоты с 3 двойными связями. В хлоропластах растений 18:3 ферментная активность, катализирующая перевод фосфатидата в диацилглицерин и диацилглицерина в моногалактозилдиацилглицерин (MGD), существенно менее выражена, чем в хлоропластах 16:3. В листьях растений 18:3 хлоропласты синтезируют стеароил-ACP2 в строме, вводят первую двойную связь в насыщенную углеводородную цепь, а затем гидролизуют тиоэфир тиоэстеразами (Фигура 1). Высвобождающийся олеат экспортируется сквозь оболочки хлоропластов в мембраны клетки, вероятно, эндоплазматический ретикулум, где он встраивается в PC. В этих мембранах в PC-связанные олеоильные группы вводятся кратные связи, и далее они перемещаются обратно в хлоропласт. MGD-связанные ацильные группы представляет собой субстраты для введения третьей двойной связи с образованием MGD с двумя линоленоильными остатками. Этот галактолипид является характерным для растений 18:3, например, таких как Asteraceae и Fabaceae. В фотосинтетически активных клетках растений 16:3, которые представлены, например, членами Apiaceae и Brassicaceae, два пути действуют параллельно, чтобы обеспечить тилакоиды MGD.
В одном варианте осуществления, растение или его часть, такой как вегетативная часть растения, по изобретению, продуцирует более высокие уровни неполярных липидов, таких как TAG, или имеет более высокое содержание общих жирных кислот (TFA), предпочтительно, оба, чем соответствующее растение или его часть, такая как вегетативная часть растения, лишенные генетических модификаций или экзогенных полинуклеотидов. В одном примере растения по изобретению образуют семена, листья или имеют части листьев с площадью поверхности по меньшей мере 1 см2, стволы и/или клубни с повышенным содержанием неполярных липидов, например, содержанием TAG или TFA, предпочтительно, обоими, по сравнению с соответствующими семенами, листьями, частями листьев с площадью поверхности по меньшей мере 1 см2, стеблями или клубнями.
В другом варианте осуществления, растение или его часть, такая как вегетативная часть растения, продуцирует TAG, обогащенные одной или несколькими конкретными жирными кислотами. Широкий спектр жирных кислот может быть включен в TAG, включая насыщенные и ненасыщенная жирные кислоты, и короткоцепочечные и длинноцепочечные жирные кислоты. Некоторые неограничивающие примеры жирных кислот, которые могут быть включены в TAG, и уровень которых может быть повышен, включают: каприновую (10:0), лауриновую (12:0), миристиновую (14:0), пальмитиновую (16:0), пальмитолеиновую (16:1), стеариновую (18:0), олеиновую (18:1), вакценовую (18:1), линолевую (18:2), элеостеариновую (18:3), γ-линоленовую (18:3), α-линоленовую (18:3ω3), стеаридоновую (18:4ω3), арахидоновую (20:0), эйкозадиеновую (20:2), дигомо-γ-линолевую (20:3), эйкозатетраеновую (20:3), арахидоновую (20:4), эйкозатетраеновую (20:4), эйкозапентаеновую (20:5ω3), бегеновую (22:0), докозапентаеновую (22:5ω), докозагексаеновую (22:6ω3) (22:63), лигноцериновую (24:0), ацетэруковую (24:1), церотиновую (26:0), и монтановую (28:0) жирные кислоты. В одном варианте осуществления настоящего изобретения, растение или его часть обогащены TAG, содержащими олеиновую кислоту и/или сниженное содержание линоленовой кислоты (ALA), предпочтительно, по меньшей мере на 2% или по меньшей мере на 5% в пересчете на абсолютное количество вещества.
Предпочтительно, растение или его часть, такая как вегетативная часть растения, по изобретению, трансформированы одним или несколькими экзогенными полинуклеотидами, такими как химерные ДНК. В случае нескольких химерных ДНК, они предпочтительно, ковалентно соединены в одну молекулу ДНК, например, такую как, единая молекула T-ДНК, и предпочтительно, интегрированы в один локус генома клетки-хозяина, предпочтительно, ядерный геном хозяина. Альтернативно, химерные ДНК находятся на двух или более молекулах ДНК, которые могут являться несвязанными в геноме хозяина, или молекула(молекулы) ДНК не являются интегрированными в геном хозяина, как это происходит в экспериментах с временной экспрессией. Растение или его часть, такой как вегетативная часть растения, предпочтительно, являются гомозиготными по одной молекуле ДНК, вставленной в их геном.
Факторы транскрипции
В клетках растений различные факторы транскрипции вовлечены в синтез жирных кислот и липидов, содержащих жирные кислоты, такие как TAG, и, таким образом, существует возможность манипулировать ими с целью модификации Push. Предпочтительным фактором транскрипции является WRI1. В настоящем описании термин «Wrinkled 1» или «WRI1» или «WRL1» обозначает фактор транскрипции класса AP2/ERWEBP, который регулирует экспрессию нескольких ферментов, принимающих участие в гликолизе и биосинтезе жирных кислот de novo. WRI1 содержит два специфических для растения (AP2/EREB) домена связывания с ДНК. Дополнительно, WRI1, по меньшей мере в Arabidopsis, также регулирует разложение сахарозы посредством гликолиза, таким образом регулируя поступление предшественников для биосинтеза жирных кислот. Иными словами, он управляет потоком углерода от продукта фотосинтеза к запасным липидам. Мутанты wri1, по меньшей мере в Arabidopsis, демонстрируют фенотип сморщенного семени, за счет дефекта во встраивании сахарозы и глюкозы в TAG.
Примеры генов, которые транскрибируются WRI1, включают, но без ограничения, одну или более, предпочтительно, все из генов, кодирующих пируваткиназу (At5g52920, At3g22960), субъединицу E1 альфа (At1g01090) пируватдегидрогеназы (PDH), ацетил-CoA карбоксилазу (ACCазу), субъединицу BCCP2 (At5g15530), еноил-ACP редуктазу (At2g05990; EAR), фосфоглицерат мутазу (At1g22170), цитозольную фруктокиназу и цитозольную фосфоглицерат мутазу, синтетазу сахарозы (SuSy) (см., например, Liu et al., 2010; Baud et al., 2007; Ruuska et al., 2002).
WRI1 содержит консервативный домен AP2 (cd00018). AP2 представляет собой домен связывания с ДНК, обнаруженный в регуляторах транскрипции в растениях, например, APETALA2 и EREBP (белок, связывающийся с элементом, который реагирует на этилен). В EREBP домен специфически связывается с боксом 11bp GCC элемента, который реагирует на этилен (ERЭ), промоторного элемента, незаменимого для реагирования на этилен. EREBP и фактор связывания с C-повтором CBF1, который принимает участие в ответе на стресс, содержат одну копию домена AP2. APETALA2-подобные белки, которые играют роль в развитии растения, содержат две копии.
Другие мотивы последовательности, которые могут быть обнаружены в WRI1 и его функциональных гомологах, включают:
1. R G V T/S R H R W T G R (SEQ ID NO:89).
2. F/Y E A H L W D K (SEQ ID NO:90).
3. D L A A L K Y W G (SEQ ID NO:91).
4. S X G F S/A R G X (SEQ ID NO:92).
5. H H H/Q N G R/K W E A R I G R/K V (SEQ ID NO:93).
6. Q E E A A A X Y D (SEQ ID NO:94).
В настоящем описании термин «Wrinkled 1» или «WRI1» также включает «подобные Wrinkled 1» или «WRI1-подобные» белки. Примеры белков WRI1 включают номера доступа: Q6X5Y6, (Arabidopsis thaliana; SEQ ID NO:22), XP_002876251.1 (подвид Arabidopsis lyrata ; Lyrata; SEQ ID NO:23), ABD16282.1 (Brassica napus; SEQ ID NO:24), ADO16346.1 (Brassica napus; SEQ ID NO:25), XP_003530370.1 (Glycine max; SEQ ID NO:26), AEO22131.1 (Jatropha curcas; SEQ ID NO:27), XP_002525305.1 (Ricinus communis; SEQ ID NO:28), XP_002316459.1 (Populus trichocarpa; SEQ ID NO:29), CBI29147.3 (Vitis vinifera; SEQ ID NO:30), XP_003578997.1 (Brachypodium distachyon; SEQ ID NO:31), BAJ86627.1 (Hordeum vulgare подвид. vulgare; SEQ ID NO:32), EAY79792.1 (Oryza sativa; SEQ ID NO:33), XP_002450194.1 (Sorghum bicolor; SEQ ID NO:34), ACG32367.1 (Zea mays; SEQ ID NO:35), XP_003561189.1 (Brachypodium distachyon; SEQ ID NO:36), ABL85061.1 (Brachypodium sylvaticum; SEQ ID NO:37), BAD68417.1 (Oryza sativa; SEQ ID NO:38), XP_002437819.1 (Sorghum bicolor; SEQ ID NO:39), XP_002441444.1 (Sorghum bicolor; SEQ ID NO:40), XP_003530686.1 (Glycine max; SEQ ID NO:41), XP_003553203.1 (Glycine max; SEQ ID NO:42), XP_002315794.1 (Populus trichocarpa; SEQ ID NO:43), XP_002270149.1 (Vitis vinifera; SEQ ID NO:44), XP_003533548.1 (Glycine max; SEQ ID NO:45), XP_003551723.1 (Glycine max; SEQ ID NO:46), XP_003621117.1 (Medicago truncatula; SEQ ID NO:47), XP_002323836.1 (Populus trichocarpa; SEQ ID NO:48), XP_002517474.1 (Ricinus communis; SEQ ID NO:49), CAN79925.1 (Vitis vinifera; SEQ ID NO:50), XP_003572236.1 (Brachypodium distachyon; SEQ ID NO:51), BAD10030.1 (Oryza sativa; SEQ ID NO:52), XP_002444429.1 (Sorghum bicolor; SEQ ID NO:53), NP_001170359.1 (Zea mays; SEQ ID NO:54), XP_002889265.1 (Arabidopsis lyrata подвид lyrata; SEQ ID NO:55), AAF68121.1 (Arabidopsis thaliana; SEQ ID NO:56), NP_178088.2 (Arabidopsis thaliana; SEQ ID NO:57), XP_002890145.1 (Arabidopsis lyrata подвид lyrata; SEQ ID NO:58), BAJ33872.1 (Thellungiella halophila; SEQ ID NO:59), NP_563990.1 (Arabidopsis thaliana; SEQ ID NO:60), XP_003530350.1 (Glycine max; SEQ ID NO:61), XP_003578142.1 (Brachypodium distachyon; SEQ ID NO:62), EAZ09147.1 (Oryza sativa; SEQ ID NO:63), XP_002460236.1 (Sorghum bicolor; SEQ ID NO:64), NP_001146338.1 (Zea mays; SEQ ID NO:65), XP_003519167.1 (Glycine max; SEQ ID NO:66), XP_003550676.1 (Glycine max; SEQ ID NO:67), XP_003610261.1 (Medicago truncatula; SEQ ID NO:68), XP_003524030.1 (Glycine max; SEQ ID NO:69), XP_003525949.1 (Glycine max; SEQ ID NO:70), XP_002325111.1 (Populus trichocarpa; SEQ ID NO:71), CBI36586.3 (Vitis vinifera; SEQ ID NO:72), XP_002273046.2 (Vitis vinifera; SEQ ID NO:73), XP_002303866.1 (Populus trichocarpa; SEQ ID NO:74), и CBI25261.3 (Vitis vinifera; SEQ ID NO:75). Дальнейшие примеры включают Sorbi-WRL1 (SEQ ID NO: 76), Lupan-WRL1 (SEQ ID NO: 77), Ricco-WRL1 (SEQ ID NO: 78), и Lupin angustifolius WRI1 (SEQ ID NO: 79). Предпочтительным WRI1 является WRI1 маиса или WRI1 сорго.
Позже, подмножество WRI1-подобных факторов транскрипции было повторно классифицировано как факторы транскрипции WRI2, WRI3 или WRI4, которые скорее отличаются преимущественной экспрессией в стеблях и/или корнях растений, чем в развивающихся семенах (To et al., 2012). Несмотря на повторную классификацию, они входят в определение «WRI1» в настоящем описании. Предпочтительными WRI1-подобными факторами транскрипции являются те, которые могут дополнить функцию мутации wri1 в растении, особенно функцию в развивающемся семени растения, например, в мутанте wri1 A. thaliana. Кроме того, функция WRI1-подобного полипептида может быть количественно оценена в N. benthamiana при помощи анализов с временной экспрессией, как описано в настоящем описании.
Фактор транскрипции WRI1 может являться эндогенным для растения или клетки, или экзогенным для растения или клетки, например, экспрессированным с экзогенного полинуклеотида. Фактор транскрипции WRI1 может представлять собой природный полипептид WRI1 или его вариант, при условии, что он сохраняет активность фактора транскрипции. Уровень или активность эндогенного полипептида WRI1 можно также увеличивать посредством увеличенной экспрессии полипептида MED15 (Kim et al., 2016), например, полипептидов, аминокислотные последовательности которых представлены как SEQ ID NO:293 или 295, или аминокислотные последовательности полипептида 14-3-3 (Ma et al., 2016), например, SEQ ID NO:297-304. Считают, что полипептид MED15 способствует нацеливанию WRI1 на его промоторы-мишени и экспрессии самого WRI1, в то время как полипептиды 14-3-3 считают взаимодействующими с полипептидом WRI1 для увеличения эффекта WRI1.
В настоящем описании полипептид «листовой семядоли» или «LEC» обозначает фактор транскрипции, который является фактором транскрипции LEC1, LEC1-подобным, LEC2, ABI3 или FUS3 и демонстрирует широкий контроль над созреванием семени и синтезом жирных кислот. LEC2, FUS3 и ABI3 представляют собой родственные полипептиды, каждый из которых содержит ДНК-связывающий домен B3 размером 120 аминокислот (Yamasaki et al., 2004), обнаруженный только в растительных белках. Их можно отличить при помощи филогенетического анализа, чтобы определить связь аминокислотной последовательности с членами множества полипептидов A. thaliana, имеющими указанные ниже номера доступа: LEC2, номер доступа AAL12004.1; FUS3 (также известный как FUSCA3), номер доступа AAC35247. LEC1 принадлежит к другому классу полипептидов и гомологичен полипептиду HAP3 класса фактора связывания CBF (Lee et al., 2003). Гены LEC1, LEC2 и FUS3 требуются в ходе раннего эмбриогенеза, чтобы предопределить гибель эмбриональных клеток и определить идентичность семядоли, и позже в ходе инициации и поддержания созревания эмбриона (Santos-Mendoza et al., 2008). Кроме того, они индуцируют экспрессию генов, кодирующих запасные белки семени, посредством связывания с мотивами RY, присутствующими в промоторах, и генов олеозина. Дополнительно, они могут отличаться характером экспрессии в ходе развития семени или своей способностью дополнить соответствующую мутацию в A. thaliana.
В настоящем описании термин «листовая семядоля 1» или «LEC1» обозначает фактор транскрипции NF-YB-типа, который принимает участие в развитии зиготы и соматическом эмбриогенезе. Эндогенный ген специфически экспрессируется в семени, как в эмбрионе, так и эндосперме. LEC1 активизирует ген, кодирующий WRI1, а также большой класс генов синтеза жирных кислот. Кроме того, эктопическая экспрессия LEC2 вызывает быструю активацию отвечающих на ауксин генов и может вызывать образование соматических эмбрионов. Примеры полипептидов LEC1 включают белки Arabidopsis thaliana (AAC39488, SEQ ID NO:149), Medicago truncatula (AFK49653, SEQ ID NO:154) and Brassica napus (ADF81045, SEQ ID NO:151), A. lyrata (XP_002862657, SEQ ID NO:150), R. communis (XP_002522740, SEQ ID NO:152), G. max (XP_006582823, SEQ ID NO:153), A. hypogaea (ADC33213, SEQ ID NO:156), Z. mays (AAK95562, SEQ ID NO:155).
LEC1-подобный (L1L) является близкородственным LEC1, но отличается другим характером экспрессии гена, который экспрессируется раньше в ходе эмбриогенеза (Kwong et al., 2003). Примеры LEC1-подобных полипептидов включают белки Arabidopsis thaliana (AAN15924, SEQ ID NO:157), Brassica napus (AHI94922, SEQ ID NO:158), and Phaseolus coccineus LEC1-подобного (AAN01148, SEQ ID NO: 159).
В настоящем описании термин «листовая семядоля 2» или «LEC2» обозначает домен B3 фактора транскрипции, который принимает участие в развитии зиготы и соматическом эмбриогенезе и который активизирует экспрессию гена, кодирующего WRI1. Его эктопическая экспрессия облегчает эмбриогенез из вегетативных тканей растения (Alemanno et al., 2008). Примеры полипептидов LEC2 включают белки Arabidopsis thaliana (номер доступа NP_564304.1, SEQ ID NO:142), Medicago truncatula (номер доступа CAA42938.1, SEQ ID NO:143) and Brassica napus (номер доступа ADO16343.1, SEQ ID NO:144).
В одном варианте осуществления, экзогенный полинуклеотид по изобретению, который кодирует LEC2, содержит одно или несколько из следующего:
i) нуклеотидов, кодирующих полипептид, содержащий аминокислоты, последовательность которых приведена в любой из SEQ ID NO: 142-144, или его биологически активный фрагмент или полипептид, аминокислотная последовательность которого по меньшей мере на 30% идентична любой одной или больше из SEQ ID NO: 142-144,
ii) нуклеотидов, последовательность которых по меньшей мере на 30% идентична i), и
iii) полинуклеотида, который гибридизуется с одним или обоими из i) или ii) в строгих условиях.
В настоящем описании термин «FUS3» обозначает домен B3 фактора транскрипции, который принимает участие в развитии зиготы и соматическом эмбриогенезе и обнаружен по большей части в протодермальной ткани эмбриона (Gazzarrini et al., 2004). Примеры полипептидов FUS3 включают белки Arabidopsis thaliana (AAC35247, SEQ ID NO:160), Brassica napus (XP_006293066.1, SEQ ID NO:161) and Medicago truncatula (XP_003624470, SEQ ID NO:162). Сверхэкспрессией любого из LEC1, L1L, LEC2, FUS3 и ABI3 из экзогенного полинуклеотида предпочтительно, управляют с помощью регулируемого развитием промотора, такого как специфический для старения промотор, индуцибельный промотор, или промотор, который сконструирован с целью обеспечения сниженного уровня экспрессии относительно природного промотора, особенно в растениях, кроме Arabidopsis thaliana и B. napus сорт Westar, во избежание аномалий развития, возникающих в ходе развития растения, которые обычно ассоциируются с повышенной экспрессией этих факторов транскрипции (Mu et al., 2008).
В настоящем описании термин «BABY BOOM» или «BBM» обозначает фактор транскрипции AP2/ERF, который индуцирует регенерацию в условиях культуры, в норме не поддерживающий регенерацию в растениях дикого типа. Эктопическая экспрессия генов Brassica napus BBM (BnBBM) в B. napus и Arabidopsis индуцирует спонтанный соматический эмбриогенез и органогенез из саженцев, выращиваемых на свободной от гормона основной среде (Boutilier et al., 2002). В табаке эктопическая экспрессия BBM является достаточной для индукции дополнительной регенерации побегов и корней на основной среде, но необходим экзогенный цитокинин для образования соматического зародыша (SE) (Srinivasan et al., 2007). Примеры полипептидов BBM включают белки Arabidopsis thaliana (номер доступа NP_197245.2, SEQ ID NO: 145), маиса (US 7579529), Sorghum bicolor (номер доступа XP_002458927) и Medicago truncatula (номер доступа AAW82334.1, SEQ ID NO:146).
В одном варианте осуществления, экзогенный полинуклеотид по изобретению, который кодирует BBM, содержит, если не указано иное, одно или несколько из следующего:
i) нуклеотидов, кодирующих полипептид, содержащий аминокислоты, последовательность которых приведена в одной из SEQ ID NO: 145 или 146, или его биологически активный фрагмент или полипептид, аминокислотная последовательность которого по меньшей мере на 30% идентична одной или обеим из SEQ ID NO: 145 или 146,
ii) нуклеотидов, последовательность которых по меньшей мере на 30% идентична i), и
iii) полинуклеотида, который гибридизуется с одним или обоими из i) или ii) в строгих условиях.
Полипептид ABI3 (A. thaliana, номер доступа NP_189108), родственный белку VP1 маиса, экспрессируется с низкими уровнями в вегетативных тканях и влияет на развитие пластид. Полипептид ABI4 A. thaliana, номер доступа NP_181551) принадлежит к семейству факторов транскрипции, которые содержат специфический для растений домен AP2 (Finkelstein et al., 1998) и действуют ниже ABI3 в биохимическом пути. ABI5 (A. thaliana номер доступа NP_565840) представляет собой фактор транскрипции семейства bZIP, который влияет на чувствительность ABA и управляет экспрессией некоторых генов LEA в семенах. Он связывает с отвечающим на ABA элементом.
Каждый из следующих факторов транскрипции был выбран на основе того, что они функционируют в ходе эмбриогенеза в растениях. Номера доступа приведены в Таблице 26. Гомологи каждого из них можно легко идентифицировать во многих другие видах растений и проанализированы, как описано в Примере 9.
MYB73 представляет собой фактор транскрипции, идентифицированный в сое, который принимает участие в стрессовых реакциях.
bZIP53 представляет собой фактор транскрипции в семействе белка bZIP, идентифицированный в Arabidopsis.
AGL15 (бесполо-подобный 15) представляет собой фактор транскрипции бокса MADS, который нативно экспрессируется в ходе эмбриогенеза. Кроме того, AGL15 нативно экспрессируется в листовом примордии, верхушечном меристеме побегов и молодых цветочных почках, наводя на мысль о том, что AGL15 может дополнительно функционировать в ходе развития после прорастания. AGL15 играет роль в эмбриогенезе и катаболизме гиббереловой кислоты. Он нацелен на домен B3 факторов транскрипции, которые являются ключевыми регуляторами эмбриогенеза.
MYB115 и MYB118 представляют собой факторы транскрипции в семействе MYB из Arabidopsis, принимающем участие в эмбриогенезе.
TANMEI, также известный как EMB2757, кодирует белок повтора WD, необходимый Arabidopsis для эмбрионального развития.
WUS, также известный как Wuschel, представляет собой ген гомеобокса, который управляет пулом стволовых клеток в эмбрионах. Он экспрессируется в стволовой клетке, организующей центр меристема, и необходим для удерживания стволовых клеток в недифференцированном состоянии. Фактор транскрипции связывается с мотивом TAAT компонента ядра.
GFR2a1 и GFR2a2 представляет собой факторы транскрипции по меньшей мере из сои.
Ацилтрансферазы жирных кислот
В настоящем описании термин «ацилтрансфераза жирных кислот» обозначает белок, способный к переносу ацильной группы с ацил-CoA, PC или ацил-ACP, предпочтительно, ацил-CoA или PC, на субстрат, с целью образования TAG, DAG или MAG. Указанные ацилтрансферазы включают DGAT, PDAT, MGAT, GPAT и LPAAT.
В настоящем описании термин «диацилглицерин-ацилтрансфераза» (DGAT) обозначает белок, который переносит жирную ацильную группу из ацил-CoA к субстрату DAG, с образованием TAG. Таким образом, термин «активность диацилглицерин-ацилтрансферазы» обозначает перенос группы ацила от ацил-CoA к DAG, с образованием TAG. DGAT может дополнительно выполнять функцию MGAT, но, в основном, функционирует, как DGAT, т. е., обладает более выраженной каталитической активностью в роли DGAT, чем в роли MGAT, если активность фермента выражена в единицах нмоль продукта/мин/мг белка (см. например, Yen et al., 2005). Активность DGAT может быть лимитирующей скорость в процессе синтеза TAG в семенах (Ichihara et al., 1988). DGAT использует субстрат ацил-CoA в качестве донора ацила и переносит его в положение sn-3 DAG, с образованием TAG. Фермент в своем нативном состоянии функционирует в эндоплазматическом ретикулуме (ER) клетки.
Существует три известных типа DGAT, обозначаемых, как DGAT1, DGAT2 и DGAT3, соответственно. Полипептиды DGAT1 являются мембранными белками, которые обычно содержат 10 трансмембранных доменов, полипептиды DGAT2 также являются мембранными белками, но обычно содержат 2 трансмембранных домена, тогда как полипептиды DGAT3 обычно не содержат ни одного и считаются растворимыми в цитоплазме, а не интегрированными в мембраны. Растительные полипептиды DGAT1 обычно содержат приблизительно 510-550 остатков аминокислот, в то время как полипептиды DGAT2 обычно содержат приблизительно 310-330 остатков. DGAT1 является основным ферментом, ответственным за продукцию TAG из DAG в большинстве развивающихся семян растений, тогда как DGAT2 из таких видов растений, как тунговое дерево (Vernicia fordii) и клещевина (Ricinus communis), которые продуцируют высокие количества необычных жирных кислот, по-видимому, играют важную роль в аккумуляции необычных жирных кислот в TAG. Повышенная экспрессия AtDGAT1 в листьях табака приводила к увеличению содержания TAG в 6-7 раз (Bouvier-Nave et al., 2000).
Примеры полипептидов DGAT1 включают белки DGAT1 Aspergillus fumigatus (XP_755172.1; SEQ ID NO:80), Arabidopsis thaliana (CAB44774.1; SEQ ID NO:1), Ricinus communis (AAR11479.1; SEQ ID NO:81), Vernicia fordii (ABC94472.1; SEQ ID NO:82), Vernonia galamensis (ABV21945.1 и ABV21946.1; SEQ ID NO:83 и SEQ ID NO:84, соответственно), Euonymus alatus (AAV31083.1; SEQ ID NO:85), Nannochloropsis oceanica (Zienkiewicz et al 2017), дрожжей (Zulu et al 2017), Caenorhabditis elegans (AAF82410.1; SEQ ID NO:86), Rattus norvegicus (NP_445889.1; SEQ ID NO:87), Homo sapiens (NP_036211.2; SEQ ID NO:88), а также их варианты и/или мутанты. Примеры полипептидов DGAT2 включают белки, кодируемые генами DGAT2 Arabidopsis thaliana (NP_566952.1; SEQ ID NO: 2), Ricinus communis (AAY16324.1; SEQ ID NO: 3), Vernicia fordii (ABC94474.1; SEQ ID NO: 4), Mortierella ramanniana (AAK84179.1; SEQ ID NO: 5), Homo sapiens (Q96PD7.2; SEQ ID NO: 6) (Q58HT5.1; SEQ ID NO: 7), Bos taurus (Q70VZ8.1; SEQ ID NO: 8), Mus musculus (AAK84175.1; SEQ ID NO: 9), а также их варианты и/или мутанты. Аминокислотные последовательности DGAT1 и DGAT2 демонстрируют незначительную гомологию. Экспрессия в листьях экзогенного DGAT2 была в два раза более эффективной, чем DGAT1 с точки зрения повышения содержания масла (TAG). Кроме того, DGAT2 A. thaliana проявляет большее предпочтение в отношении линолеоил-CoA и линоленоил-CoA в качестве доноров ацила, по сравнению с олеоил-CoA, сравнению с DGAT1. Указанное предпочтение в отношении субстрата может применяться, чтобы отличить два класса DGAT, в добавление к их аминокислотным последовательностям.
Примеры полипептидов DGAT3 включают белки, кодируемые генами DGAT3 арахиса (Arachis hypogaea, Saha et al., 2006), а также их вариантами и/или мутантами. DGAT обладает незначительной или не обладает обнаружимой активностью MGAT, например, менее 300 пмоль/мин/мг белка, предпочтительно, менее 200 пмоль/мин/мг белка, более предпочтительно, менее 100 пмоль/мин/мг белка.
В одном варианте осуществления, экзогенный полинуклеотид по изобретению, кодирующий DGAT1, содержит одно или больше из следующего:
i) нуклеотидов, кодирующих полипептид, содержащий аминокислоты, последовательность которых приведена в любой из SEQ ID NO: 1 или 80-88, или его биологически активный фрагмент или полипептид, последовательность аминокислот которого по меньшей мере на 30% идентична любой одной или более из SEQ ID NO: 1 или 80-88,
ii) нуклеотидов, последовательность которых по меньшей мере на 30% идентична i), и
iii) полинуклеотида, который гибридизуется с одним или обоими из i) или ii) в строгих условиях.
В одном варианте осуществления, экзогенный полинуклеотид по изобретению, кодирующий DGAT2, содержит одно или несколько из следующего
i) нуклеотидов, кодирующих полипептид, содержащий аминокислоты, последовательность которых приведена в любой из SEQ ID NO: 2-9, или его биологически активный фрагмент или полипептид, последовательность аминокислот которого по меньшей мере на 30% идентична любой одной или больше из SEQ ID NO: 2-9,
ii) нуклеотидов, последовательность которых по меньшей мере на 30% идентична i), и
iii) полинуклеотида, который гибридизуется с одним или обоими из i) или ii) в строгих условиях.
Как используют в настоящем описании, термин «фосфолипид:диацилглицерин-ацилтрансфераза» (PDAT; EC 2.3.1.158) или его синоним «фосфолипид:1,2-диацил-sn-глицерид-O-ацилтрансфераза» обозначает ацилтрансферазу, переносящую ацильную группу с фосфолипида, как правило, с положения sn-2 PC в положение sn-3 DAG, с образованием TAG и лизофосфохолина (LPC). Эта реакция отличается от реакции DGAT и в ней используются фосфолипиды в качестве доноров ацила. Увеличенная экспрессия PDAT, такого как PDAT1, который может являться экзогенным или эндогенным для клетки или растения по изобретению, увеличивает продукцию TAG из PC. Фермент LPCAT может повторно ацилировать LPC для образования большего количества PC, позволяя непрерывную продукцию DAG посредством PDAT. Существует несколько форм PDAT в клетках растений, включая PDAT1, PDAT2 или PDAT3 (Ghosal et al., 2007). Последовательности иллюстративных кодирующих областей и полипептидов PDAT представлены в настоящем описании как SEQ ID NO:258-261 (PDAT1 Sorghum и Zea mays, No доступа XM_002462417.1 и NM_001147943), (Dahlqvist et al., 2000; Fan et al., 2013; Fan et al., 2014), хотя можно использовать любой ген, кодирующий PDAT. Гомологи и природные варианты PDAT их этих или других видов растений, грибов или водорослей можно легко идентифицировать и использовать по настоящему изобретению. В одном варианте осуществления, гомолог или вариант является по меньшей мере на 95% идентичным, предпочтительно, по меньшей мере на 99% идентичным, аминокислотной последовательности из перечисленных SEQ ID NO или No. доступа. PDAT может являться экзогенным или эндогенным для растения или его части.
В настоящем описании термин «моноацилглицерин-ацилтрансфераза» или «MGAT» обозначает белок, который переносит жирную ацильную группу из ацил-CoA к субстрату MAG, например, sn-2 MAG, для образования DAG. Таким образом, термин «активность моноацилглицерин-ацилтрансферазы» по меньшей мере обозначает перенос ацильной группы от ацил-CoA к MAG, для образования DAG. Термин «MGAT» в настоящем описании включает ферменты, которые действуют на субстраты sn-1/3 MAG и/или sn-2 MAG с образованием sn-1,3 DAG и/или sn-1,2/2,3-DAG, соответственно. В предпочтительном варианте осуществления MGAT отдает предпочтение субстрату sn-2 MAG относительно sn-1 MAG, или в основном использует в качестве субстрата только sn-2 MAG. В настоящем описании MGAT не включает ферментов, которые предпочтительно, переносят ацильную группу к LysoPA относительно MAG, такие ферменты известны как LPAAT. Таким образом, MGAT предпочтительно, использует нефосфорилированные моноацильные субстраты, даже если они также могут обладать низкой каталитической активностью по отношению к LysoPA. Предпочтительный MGAT не обладает обнаружимой активностью ацилирования LysoPA. Кроме того, MGAT может выполнять функцию DGAT, но, в основном, функционирует как MGAT, т. е., он обладает более выраженной каталитической активностью в роли MGAT, чем в роли DGAT, если активность фермента выражается в единицах нмоль продукта/мин/мг белка (также см. Yen et al., 2002). Существует три известных класса MGAT, обозначаемых, как MGAT1, MGAT2 и MGAT3, соответственно. Примеры полипептидов MGAT1, MGAT2 и MGAT3 описаны в WO2013/096993.
В настоящем описании «путь MGAT» обозначает анаболический путь образования TAG, отличный от пути Кеннеди, в котором DAG образуется ацилированием sn-1 MAG или, предпочтительно, sn-2 MAG, катализированным MGAT. В дальнейшем DAG может быть использован для образования TAG или других липидов. В WO2012/000026 продемонстрировано, во-первых, что листовая ткань растения может синтезировать MAG из G-3-P таким образом, что MAG доступен экзогенной MGAT, экспрессирующейся в ткани листа, во-вторых, что MGAT из различных источников может функционировать в тканях растения, что требует успешного взаимодействия с другими факторами растения, принимающими участие в синтезе липидов, и в-третьих, что DAG, образованный под действием экзогенной MGAT, доступен растительной DGAT или экзогенной DGAT для продукции TAG. Активность MGAT и DGAT может быть проанализирована посредством введения конструкций, кодирующих ферменты (или ферменты-кандидаты) в Saccharomyces cerevisiae, штамм H1246 и демонстрации накопления TAG.
Некоторые из мотивов, важность которых для каталитической активности DGAT2 продемонстрирована в некоторых DGAT2, также являются консервативными в MGAT ацилтрансферазах. Особый интерес представляет предполагаемый домен связывания с нейтральными липидами с консенсусной последовательностью FLXLXXXN (SEQ ID NO: 14), где каждый X независимо представляет собой любую аминокислоту, кроме пролина, и N представляет собой любую неполярную аминокислоту, расположенную в пределах N-концевого трансмембранного участка, после которого следует предполагаемый домен глицерил/фосфолипид ацилтрансферазы. Мотив FLXLXXXN (SEQ ID NO: 14) обнаружен в DGAT2 мыши (аминокислоты 81-88) и MGAT1/2, но не в DGAT2 дрожжей или растения. Он является важным для активности DGAT2 мыши. Другие мотивы последовательности DGAT2 и/или MGAT1/2 включают:
1. В высокой степени консервативный трипептид YFP (SEQ ID NO: 10) в большинстве полипептидов DGAT2, а также в MGAT1 и MGAT2, например, присутствующий как аминокислоты 139-141 в DGAT2 мыши. Мутация данного мотива в DGAT2 дрожжей посредством неконсервативных замен делает фермент нефункциональным.
2. Тетрапептид HPHG (SEQ ID NO: 11), в высокой степени консервативный в MGAT, а также в последовательностях DGAT2 животных и грибов, например, присутствующий как аминокислоты 161-164 в DGAT2 мыши, и важный для каталитической активности по меньшей мере в DGAT2 дрожжей и мыши. Растительные ацилтрансферазы DGAT2 содержат вместо него консервативную последовательность EPHS (SEQ ID NO: 12), поэтому консервативные модификации в первом и четвертом положениях аминокислот могут быть переносимыми.
3. Более длинный консервативный мотив, который является частью предполагаемого домена фосфолипида глицерина. Примером данного мотива является
RXGFX(K/R)XAXXXGXXX(L/V)VPXXXFG(E/Q) (SEQ ID NO:13), который присутствует как аминокислоты 304-327 в DGAT2 мыши. Последовательность аминокислот в данном мотиве менее консервативна, чем в остальных, как можно было бы ожидать на основании его длины, и гомологи могут быть распознаны посредством поиска мотива. Расположение может варьировать для более консервативных аминокислот, т. е., могут присутствовать дополнительные X аминокислоты в пределах мотива или меньшее количество X аминокислот, по сравнению с приведенной выше последовательностью.
Одним важным компонентом в синтезе глицеролипида из жирных кислот, этерифицированных ACP или CoA, является фермент, sn-глицерил-3-фосфат ацилтрансфераза (GPAT), которая представляет собой другой полипептид, вовлеченный в биосинтез неполярных липидов. Указанный фермент принимает участие в различных метаболических путях и осуществлении физиологических функций. Он катализирует следующую реакцию: G3P+жирный ацил-ACP или -CoA → LPA+свободный-ACP или -CoA. Катализируемая GPAT реакция происходит в трех отдельных субклеточных структурах растения: пластиде, эндоплазматическом ретикулуме (ER) и митохондрии. Указанные реакции катализируются тремя различными видами ферментов GPAT, растворимой формой, которая локализована в строме пластиды, которая использует ацил-ACP в качестве своего природного ацильного субстрата (PGPAT на Фигура 1), и двумя мембраносвязанными формами, локализованными в ER и митохондрии, которые используют ацил-CoA и ацил-ACP в качестве природных доноров ацила, соответственно (Chen et al., 2011).
В настоящем описании термин «глицерил-3-фосфат ацилтрансфераза» («GPAT»; EC 2.3.1.15) и его синоним «глицерил-3-фосфат O-ацилтрансфераза» обозначают белок, который ацилирует глицерил-3-фосфат (G-3-P), с образованием LysoPA и/или MAG, где последний продукт образуется, если GPAT дополнительно обладает активностью фосфатазы по отношению к LysoPA. Ацильная группа, которая переносится, происходит от ацил-CoA, если GPAT относится к ER-типу GPAT («ацил-CoA:sn-глицерил-3-фосфат 1-O-ацилтрансфераза», дополнительно обозначается как «микросомальная GPAT»), или от ацил-АЦФ, если GPAT является GPAT пластидного типа (GPATПТ). Таким образом, термин «активность глицерил-3-фосфат ацилтрансферазы» обозначает ацилирование G-3-P с образованием LysoPA и/или MAG. Термин «GPAT» включает ферменты, которые ацилируют G-3-P с образованием sn-1 LPA и/или sn-2 LPA, предпочтительно, sn-2 LPA. Предпочтительно, GPAT, которая может повышенно экспрессироваться при модификации Pull, представляет собой мембраносвязанную GPAT, которая функционирует в ER клетки, более предпочтительно, GPAT9, а пластидная GPAT, которая подавляется при модификации Prokaryotic pathway, представляет собой растворимую GPAT («пластидную GPAT»).В предпочтительном варианте осуществления GPAT обладает активностью фосфатазы. В наиболее предпочтительном варианте осуществления GPAT представляет собой sn-2 GPAT, обладающий активностью фосфатазы, которая продуцирует sn-2 MAG.
В настоящем описании термин « sn-1 глицерил-3-фосфат ацилтрансфераза» (sn-1 GPAT) обозначает белок, который ацилирует sn-глицерил-3-фосфат (G-3-P), с предпочтительным образованием 1-ацил -sn-глицерил-3-фосфата (sn-1 LPA). Таким образом, термин «активность sn-1 глицерил-3-фосфат ацилтрансферазы» обозначает ацилирование sn-глицерил-3-фосфата с образованием 1-ацил-sn-глицерил-3-фосфата (sn-1 LPA).
В настоящем описании термин «sn-2 глицерил-3-фосфат ацилтрансфераза» (sn-2 GPAT) обозначает белок, который ацилирует sn-глицерил-3-фосфат (G-3-P) с предпочтительным образованием 2-ацил-sn-глицерил-3-фосфата (sn-2 LPA). Таким образом, термин «активность sn-2 глицерил-3-фосфат ацилтрансферазы» обозначает ацилирование sn-глицерил-3-фосфата с образованием 2-ацил-sn-глицерил-3-фосфата (sn-2 LPA).
Семейство GPAT велико, и все известные члены содержат два консервативных домена, ацилтрансферазный домен plsC (PF01553; SEQ ID NO: 15) и домен суперсемейства галогенкислоты дегалогеназа-подобной гидролазы (PF12710; SEQ ID NO: 16), а также их варианты. В дополнение к этому, по меньшей мере в Arabidopsis thaliana, все GPAT в подклассах GPAT4-GPAT8 содержат N-концевой участок, гомологичный домену фосфосерин фосфатазы (PF00702; SEQ ID NO: 17), и GPAT, которые продуцируют MAG в качестве продукта, могут быть идентифицированы по наличию такого гомологичного участка. Некоторые GPAT, эндогенно экспрессируемые в ткани листа, содержат консервативную последовательность аминокислот GDLVICPEGTTCREP (SEQ ID NO:18). GPAT4 и GPAT6 содержат консервативные остатки, которые известны как критические для активности фосфатазы, конкретно консервативные аминокислоты в мотиве I (DXDX[T/V][L/V]; SEQ ID NO: 19) и мотиве III (K-[G/S][D/S]XXX[D/N]; SEQ ID NO: 20), расположены на N-конце (Yang et al., 2010).
Гомологи Arabidopsis GPAT4 (номер доступа NP_171667.1) и GPAT6 (NP_181346.1) включают AAF02784.1 (Arabidopsis thaliana), AAL32544.1 (Arabidopsis thaliana), AAP03413.1 (Oryza sativa), ABK25381.1 (Picea sitchensis), ACN34546.1 (Zea Mays), BAF00762.1 (Arabidopsis thaliana), BAH00933.1 (Oryza sativa), EAY84189.1 (Oryza sativa), EAY98245.1 (Oryza sativa), EAZ21484.1 (Oryza sativa), EEC71826.1 (Oryza sativa), EEC76137.1 (Oryza sativa), EEE59882.1 (Oryza sativa), EFJ08963.1 (Selaginella moellendorffii), EFJ11200.1 (Selaginella moellendorffii), NP_001044839.1 (Oryza sativa), NP_001045668.1 (Oryza sativa), NP_001147442.1 (Zea mays), NP_001149307.1 (Zea mays), NP_001168351.1 (Zea mays), AFH02724.1 (Brassica napus) NP_191950.2 (Arabidopsis thaliana), XP_001765001.1 (Physcomitrella patens), XP_001769671.1 (Physcomitrella patens), (Vitis vinifera), XP_002275348.1 (Vitis vinifera), XP_002276032.1 (Vitis vinifera), XP_002279091.1 (Vitis vinifera), XP_002309124.1 (Populus trichocarpa), XP_002309276.1 (Populus trichocarpa), XP_002322752.1 (Populus trichocarpa), XP_002323563.1 (Populus trichocarpa), XP_002439887.1 (Sorghum bicolor), XP_002458786.1 (Sorghum bicolor), XP_002463916.1 (Sorghum bicolor), XP_002464630.1 (Sorghum bicolor), XP_002511873.1 (Ricinus communis), XP_002517438.1 (Ricinus communis), XP_002520171.1 (Ricinus communis), ACT32032.1 (Vernicia fordii), NP_001051189.1 (Oryza sativa), AFH02725.1 (Brassica napus), XP_002320138.1 (Populus trichocarpa), XP_002451377.1 (Sorghum bicolor), XP_002531350.1 (Ricinus communis), and XP_002889361.1 (Arabidopsis lyrata).
Были выделены растворимые пластидные GPAT (PGPAT, также известные как ATS1 в Arabidopsis thaliana), где кодирующие их гены клонированы из нескольких видов растений, таких как горох (Pisum sativum, номер доступа: P30706.1), шпинат (Spinacia oleraceа, номер доступа: Q43869.1), тыква (Cucurbita moschate, номер доступа: P10349.1), огурец (Cucumis sativus, номер доступа: Q39639.1) и Arabidopsis thaliana (номер доступа: Q43307.2). Растворимая пластидная GPAT представляет собой первую фиксированную стадию того, что известно как прокариотический путь синтеза глицеролипидов и действует только в пластиде (Фигура 1). Так называемый прокариотический путь расположен исключительно в пластидах растений и обеспечивает сборку DAG для синтеза галактолипидов (MGDG и DGMG), которые содержат C16:3 жирные кислоты, этерифицированные в положении sn-2 глицеринового остова.
Консервативные мотивы и/или остатки можно использовать в качестве основанного на последовательности диагностического признака для идентификации ферментов GPAT. Альтернативно, может применяться более строгий анализ, основанный на функции. Такой анализ включает, например, подкормку меченым глицерил-3-фосфатом клеток или микросом и количественное определение уровней меченых продуктов тонкослойной хроматографией или подобным способом. Активность GPAT приводит к образованию меченой LPA, тогда как активность GPAT/фосфатазы приводит к образованию меченого MAG.
В настоящем описании термин «ацилтрансфераза лизофосфатидиновой кислоты» (LPAAT; EC 2.3.1.51) и его синонимы «1-ацил-глицерил-3-фосфат ацилтрансфераза», «ацил-CoA:1-ацил-sn-глицерол-3-фосфат-2-O-ацилтрансфераза» и «1-ацилглицерил-3-фосфат O-ацилтрансфераза» обозначают белок, который ацилирует лизофосфатидиновую кислоту (LPA) с образованием фосфатидиновой кислоты (PA, PA). Ацильную группу для переноса получают из ацил-CoA, если LPAAT представляет собой LPAAT ER-типа, или из ацил-ACP, если LPAAT представляет собой LPAAT пластидного типа (ПLPAAT). Таким образом, термин «активность ацилтрансферазы лизофосфатидиновой кислоты» обозначает ацилирование LPA с образованием PA.
Полипептиды покрытия масляных телец
Семена растения и пыльца накапливают TAG в субклеточных структурах, называемых масляными тельцами, диаметр которых в общем находится в диапазоне 0,5-2,5 мкм. В настоящем описании «липидные капельки», также называемые «масляными тельцами», представляют собой богатые липидами клеточные органеллы для хранения или обмена нейтральных липидов, в основном содержащие TAG. Размер липидных капелек может значительно варьировать, от приблизительно 20 нм до 100 мкм. Указанные органеллы содержат ядро TAG, окруженное фосфолипидным монослоем, содержащим несколько инкорпорированных белков, принимающих участие в метаболизме и хранении липидов, а также в переносе липидов к другим мембранам, в том числе олеозинов, если масляные тельца происходят из растительных семян или тканей цветка (Jolivet et al., 2004). В общем, они содержат 0,5-3,5% белка, в то время как остальная часть представлена липидом. Они являются наименее плотными органеллами в большинстве клеток и могут, таким образом, с легкостью быть выделены посредством центрифугирования с флотацией. Олеозины представляют самый распространенный (по меньшей мере 80%) белок в мембране масляных телец из семян.
В одном варианте осуществления, полипептид покрытия масляных телец является неаллергенным, или неизвестным как аллергенный, например, для человека. Как используют в настоящем описании, термин «аллергенный полипептид» обозначает полипептид, отличающийся присутствием двух признаков: (i) его аминокислотная последовательность содержит область по меньшей мере из 80 смежных аминокислот, последовательность которых является по меньшей мере на 35% идентичной последовательности по меньшей мере из 80 смежных аминокислот известного аллергенного белка, и (ii) его аминокислотная последовательность содержит по меньшей мере 8 смежных аминокислот, последовательность которых является идентичной области по меньшей мере из 8 смежных аминокислот известного аллергенного белка. Как используют в настоящем описании, «неаллергенный полипептид» представляет собой полипептид, который не является аллергенным полипептидом. Предпочтительные неаллергенные полипептиды представляют собой полипептиды, не имеющие каждого из признаков (i) и (ii). Для ясности, неаллергенные полипептиды могут иметь признак (i) или (ii), но не оба (i) и (ii). Подгруппа неаллергенных полипептидов имеют признак (i), но не признак (ii); они являются менее предпочтительными, чем полипептиды, не имеющие ни признака (i), ни признака (ii).
Признаки, описанные как (i) и (ii), можно определять посредством проведения поиска с использованием базы данных AllergenOnline и механизма поиска, доступного на www.allergenonline.org. Проводят два поиска с использованием аминокислотной последовательности представляющего интерес полипептида, который используют в качестве запроса для поиска в базе данных последовательностей известных аллергенов на AllergenOnline. В первом поиске используют скользящее окно из 80 аминокислот из представляющего интерес полипептида (аминокислоты 1-80, 2-81, 3-82 и т.д.), проводя поиск совпадений по меньшей мере с 35% идентичностью посредством программы FASTA (Pearson and Lipman, 1988). 35% идентичность для фрагментов из 80 аминокислот предложена в научной консультации регулирующим органам для оценки полипептидов в генетически модифицированных сельскохозяйственных культурах, см. FAO/WHO 2001 и Codex 2003. Способ поиска совпадений фрагментов, оценивающий фрагменты из 80 аминокислот, по-видимому, является достаточно консервативным. То есть, при использовании только этого первого поиска при классификации полипептидов как потенциально аллергеных, положительное совпадение с 35% идентичностью может привести к ошибочной классификации полипептидов как потенциально аллергенных, когда степень идентичности не имеет биологической значимости для аллергенности. Таким образом, проводят также второй поиск совпадения 8 аминокислот, и представляющий интерес полипептид классифицируют как потенциальный аллерген на основании положительного совпадения в обоих поисках, не только в одном поиске.
При проведении поиска в базе данных AllergenOnline с использованием запрашиваемых последовательностей, для полипептидов, включающих WRI1 маиса, DGAT1 Arabidopsis, DGAT1.1 пальмы, GPAT9 кокоса, FatA2 Arabidopsis, калеозин Arabidopsis (At2g33380), LDSP Nannochloropsis, фибриллин перца, TadA Rhodococcus и все тестированные калеозины, не показали совпадений при поиске в скользящем окне из 80 аминокислот, и таким образом, их классифицировали как неаллергенные. Для всех других последовательностей, включая олеозин U1 ванили, LDAP2 китайского сального дерева, стеролеозин Arabidopsis (At5g50600), олеозин 3 арахиса, олеозин H кунжута, олеозин авокадо, олеозин инжира, олеозин огурца, олеозин льна, олеозин сои, олеозин Brassica и олеозин картофеля, получили одно или несколько совпадений в поиске в скользящем окне из 80 аминокислот (т.е. по меньшей мере 35% идентичность с известным аллергенным белком в области по меньшей мере из 80 аминокислот), но не получили никаких совпадений в поиске для 8 аминокислот. Их, таким образом, классифицировали как неаллергенные в соответствии с определением выше. В противоположность этому, олеозин L кунжута идентифицировали в качестве аллергенного полипептида, имеющего совпадения в обоих поиска.
В настоящем описании термин «олеозин» обозначает амфифильный белок, присутствующий в мембране масляных телец в запасных тканях семян (см., например, Huang, 1996; Tai et al., 2002, Lin et al., 2005; Capuano et al., 2007; Lui et al., 2009; Shimada и Hara-Nishimura, 2010), и искусственно созданные варианты (см., например WO2011/053169 и WO2011/127118).
Олеозины обладают низкой М.м. (15-26000), что соответствует приблизительно 140-230 аминокислотных остатков, и это позволяет им плотно упаковываться на поверхности масляных телец. В семенах каждого вида обычно представлены два или более олеозинов с различной М.м.. Каждая молекула олеозина содержит относительно гидрофильный, вариабельный N-концевой домен (например, длиной приблизительно 48 остатков аминокислоты), центральный полностью гидрофобный домен (например, длиной приблизительно 70-80 остатков аминокислоты), который особенно богат алифатическими аминокислотами, такими как аланин, глицин, лейцин, изолейцин и валин, и амфифильный α-спиральный домен размером приблизительно 30-40 остатков аминокислот на C-конце или вблизи него. Обычно центральный гидрофобный домен содержит в центре мотив пролинового узла размером приблизительно 12 остатков. В общем, центральный участок гидрофобных остатков вставлен в липидное ядро, и амфифильный N-концевой участок и/или амфифильный C-концевой участок размещен на поверхности масляных телец, где положительно заряженные остатки инкорпорированы в фосфолипидный монослой, и отрицательно заряженные участки контактируют с внешней средой.
В настоящем описании термин «олеозин» включает полиолеозины, содержащие несколько полипептидов олеозина, соединенных вместе способом «голова к хвосту» в качестве единого полипептида (WO2007/045019), например, 2x, 4x или 6x пептидов олеозина, и калеозины, которые связывают кальций и составляют минорный белковый компонент белков, покрывающих масляные тельца в семенах (Froissard et al., 2009), а также стеролеозины, которые связываются со стеролами (WO2011/053169). Однако, в общем, значительная часть (по меньшей мере 80%) олеозинов в масляных тельцах не будет относиться к калеозинам и/или стеролеозинам. Термин «олеозин» дополнительно включает полипептиды олеозина, модифицированные искусственным посредством, т.е. олеозины, в которых один или более остатков аминокислот природного олеозина искусственно заменены остатками цистеина, как описано в WO2011/053169. Обычно, 4-8 остатков заменены искусственным посредством, предпочтительно, 6 остатков, но может быть заменено от 2 до 14 остатков. Предпочтительно, амфифильные N-концевой и C-концевой домены содержат замены цистеина. Модификация повышает способность олеозинов к образованию поперечных связей и повышает термостабильность и/или устойчивость белков к разложению протеазами.
Известно значительное количество последовательностей белка олеозина и нуклеотидных последовательностей, кодирующих его, из множества различных видов растений. Примеры включают, но без ограничения, олеозины из кунжута, Arabidposis, рапса, кукурузы, риса, арахиса, клещевины, сои, льна, винограда, капусты, хлопчатника, подсолнечника, сорго и ячменя. Примеры олеозинов (с номерами доступа) включают олеозин Brassica napus (CAA57545.1; SEQ ID NO:95), Brassica napus олеозин S1-1 (ACG69504.1; SEQ ID NO:96), Brassica napus олеозин S2-1 (ACG69503.1; SEQ ID NO:97), Brassica napus олеозин S3-1 (ACG69513.1; SEQ ID NO:98), Brassica napus олеозин S4-1 (ACG69507.1; SEQ ID NO:99), Brassica napus олеозин S5-1 (ACG69511.1; SEQ ID NO:100), Arachis hypogaea олеозин 1 (AAZ20276.1; SEQ ID NO:101), Arachis hypogaea олеозин 2 (AAU21500.1; SEQ ID NO:102), Arachis hypogaea олеозин 3 (AAU21501.1; SEQ ID NO:103), Arachis hypogaea олеозин 5 (ABC96763.1; SEQ ID NO:104), Ricinus communis олеозин 1 (EEF40948.1; SEQ ID NO:105), Ricinus communis олеозин 2 (EEF51616.1; SEQ ID NO:106), олеозин Glycine max изоформа a (P29530.2; SEQ ID NO:107), олеозин Glycine max изоформа b (P29531.1; SEQ ID NO:108), олеозин Linum usitatissimum низкомолекулярная изоформа (ABB01622.1; SEQ ID NO:109), олеозин Linum usitatissimum высокомолекулярная изоформа (ABB01624.1; SEQ ID NO:110), олеозин Helianthus annuus (CAA44224.1; SEQ ID NO:111), олеозин Zea mays (NP_001105338.1; SEQ ID NO:112), стеролеозин Brassica napus (ABM30178.1; SEQ ID NO:113), Brassica napus стеролеозин SLO1-1 (ACG69522.1; SEQ ID NO:114), Brassica napus стеролеозин SLO2-1 (ACG69525.1; SEQ ID NO:115), стеролеозин Sesamum indicum (AAL13315.1; SEQ ID NO:116), Sesame indicum олеозин L (Tai et al., 2002; номер доступа AF091840; SEQ ID NO:305), Ficus pumila var. Awkeotsang изоформа олеозин L (номер доступа ABQ57397.1; SEQ ID NO: 306), Cucumis sativus олеозин L (номер доступа XP_004146901.1; SEQ ID NO: 307), Linum usitatissimum олеозин L (номер доступа ABB01618.1; SEQ ID NO: 308), Glycine max олеозин L (номер доступа XP_003556321.2; SEQ ID NO: 309), Ananas comosus олеозин L (номер доступа OAY72596.1; SEQ ID NO: 310), Setaria italica олеозин L (номер доступа XP_004956407.1; SEQ ID NO: 311), Fragaria vesca подвид vesca олеозин L (номер доступа XP_004307777.1; SEQ ID NO: 312), Brassica napus олеозин L (номер доступа CDY03377.1; SEQ ID NO: 313), Solanum lycopersicum олеозин L (номер доступа XP_004240765.1; SEQ ID NO: 314), Sesame indicum олеозин H1 (Tai et al., 2002; номер доступа AF302807), олеозин U1 листьев Vanilla planifolia (Huang and Huang, 2016; номер доступа SRX648194), ассоциированный с липидными капельками белок Persea americana мезокарпий олеозин M (Huang and Huang, 2016; номер доступа SRX627420), Arachis hypogaea олеозин 3 (Parthibane et al., 2012; номер доступа AY722696), A. thaliana калеозин3 (Shen et al., 2014; Laibach et al., 2015; номер доступа AK317039), A. thaliana стеролеозин (номер доступа AT081653), стеролеозин Zea mays (NP_001152614.1; SEQ ID NO:117), калеозин CLO-1 Brassica napus (ACG69529.1; SEQ ID NO:118), Brassica napus калеозин CLO-3 (ACG69527.1; SEQ ID NO:119), калеозин Sesamum indicum (AAF13743.1; SEQ ID NO:120), калеозин Zea mays (NP_001151906.1; SEQ ID NO:121), калеозин Glycine max (AAB71227). Другими функционально эквивалентными полипептидами липидной инкапсуляции являются пластоглобулины и полипептиды белка жировых капель миокарда (БFAМ) (WO2011/127118).
В одном варианте осуществления, экзогенный полинуклеотид по изобретению, кодирующий олеозин, содержит, если не указано иное, одно или несколько из следующего:
i) нуклеотиды, кодирующие полипептид, содержащий аминокислоты, последовательность которых приведена в любой из SEQ ID NO: 95-112 или 305-314, или его биологически активный фрагмент или полипептид, последовательность аминокислот которого по меньшей мере на 30% идентична любой одной или более из SEQ ID NO: 95-112 или 305-314,
ii) нуклеотиды, последовательность которых по меньшей мере на 30% идентична i), и
iii) полинуклеотид, который гибридизуется с одним или обоими из i) или ii) в строгих условиях.
В одном варианте осуществления, экзогенный полинуклеотид по изобретению, кодирующий стеролеозин, содержит, если не указано иное, одно или несколько из следующего:
i) нуклеотиды, кодирующие полипептид, содержащий аминокислоты, последовательность которых приведена в любой из SEQ ID NO: 113-117, или его биологически активный фрагмент или полипептид, последовательность аминокислот которого по меньшей мере на 30% идентична одной или более из SEQ ID NO: 113-117,
ii) нуклеотиды, последовательность которых по меньшей мере на 30% идентична i), и
iii) полинуклеотид, который гибридизуется с одним или обоими из i) или ii) в строгих условиях.
В одном варианте осуществления, олеозин представляет собой олеозин L или его ортолог. В олеозине L отсутствует инсерция H-формы приблизительно из 18 аминокислот на C-конце других олеозинов (см., например, Tai et al., 2002). Таким образом, олеозин L можно легко отличать от других олеозинов на основании выравнивания белка.
В одном варианте осуществления, экзогенный полинуклеотид по изобретению, кодирующий олеозин L, содержит, если не определено иное, одно или несколько из следующего:
i) нуклеотидов, кодирующих полипептид, содержащий аминокислоты, последовательность которых указана как любая из SEQ ID NO: 305-314, или его биологически активный фрагмент, или полипептид, аминокислотная последовательность которого является по меньшей мере на 30% идентичной любой одной или нескольким из SEQ ID NO: 305-314,
ii) нуклеотидов, последовательность которых является по меньшей мере на 30% идентичной i), и
iii) полинуклеотида, который гибридизуется с одним или обоими из i) или ii) в строгих условиях.
В альтернативном варианте осуществления, экзогенный полинуклеотид по изобретению, кодирующий олеозин, содержит, если не определено иное, одно или несколько из следующего:
i) нуклеотидов, кодирующих полипептид, содержащий аминокислоты, последовательность которых указана как любая из SEQ ID NO: 306-314, или его биологически активный фрагмент, или полипептид, аминокислотная последовательность которого является по меньшей мере на 30% идентичной любой одной или нескольким из SEQ ID NO: 306-314,
ii) нуклеотидов, последовательность которых является по меньшей мере на 30% идентичной i), и
iii) полинуклеотида, который гибридизуется с одним или обоими из i) или ii) в строгих условиях, где олеозин является неаллергеным, или неизвестным как аллергенный, например, для чеовека.
В настоящем описании «ассоциированный с липидными капельками белок» или «LDAP» обозначает полипептид, который в растениях связан с липидными капельками в тканях или органах, кроме семян, пыльников и пыльцы, например, тканях фруктов, включая перикарпий и мезокарпий. LDAP могут быть связаны с масляными тельцами в семенах, пыльниках или пыльце, а также в тканях или органах, кроме семян, пыльников и пыльцы. Они отличаются от олеозинов, которые являются полипептидами, связанными с поверхностью липидных капелек в тканях семени, пыльниках и пыльце. LDAP в настоящем описании включают полипептиды LDAP, образованные природным посредством в тканях растения, а также варианты аминокислотной последовательности, полученные искусственным способов. Функция таких вариантов может быть проанализирована, как проиллюстрировано в Примере 11.
Horn et al. (2013) идентифицировали два гена LDAP, которые экспрессируются в перикарпии авокадо. Аминокислотная последовательность кодируемых полипептидов авокадо LDAP1 и LDAP2 была на 62% идентичной, а также обладала гомологией к полипептиду, кодируемому At3g05500 Arabidopsis и SRPP-подобному белку резинового дерева. Gidda et al. (2013) идентифицировали три гена LDAP, которые экспрессировались в мезокарпии, но не в ядрах масличной пальмы (Elaeis guineensis) и сделали вывод о том, что гены LDAP были специфическими для растения и консервативными для всех видов растений. Полипептиды LDAP могут содержать дополнительные домены (Gidda et al., (2013). Гены, кодирующие LDAP, в общем, активизируются в тканях, не относящихся к семенам, с обильным содержанием липидов, и могут быть идентифицированы таким образом, однако считается, что они экспрессируются во всех клетках, не относящихся к семенам, которые продуцируют масло, в том числе для временного хранения. Horn et al. (2013) иллюстрируют филогенетическое дерево SRPP-подобных белков в растениях. В Примере 11 и Примере 17 настоящего описания описаны характерные полипептиды LDAP, такие как ассоциированный с липидными капельками белок TadA Rhodococcus opacus (MacEachran et al., 2010; Номер доступа HM625859), белок масляных телец LSDP Nannochloropsis oceanica (Vieler et al., 2012; Номер доступа JQ268559) и гидрофобин HFBI Trichoderma reesei (Linder et al., 2005; Номер доступа Z68124). Специалист в данной области может легко идентифицировать гомологи LDAP в других видах растений.
В одном варианте осуществления, экзогенный полинуклеотид по изобретению, кодирующий LDAP, содержит, если не указано иное, одно или несколько из следующего:
i) нуклеотидов, кодирующих полипептид, содержащий аминокислоты, последовательность которых приведена в любой из SEQ ID NO: 228, 230 или 232, или его биологически активный фрагмент или полипептид, последовательность аминокислот которого по меньшей мере на 30% идентична любой одной или более из SEQ ID NO: 228, 230 или 232,
ii) нуклеотидов, последовательность которых по меньшей мере на 30% идентична i), и
iii) полинуклеотида, который гибридизуется с одним или обоими из i) или ii) в строгих условиях.
В настоящем описании термин «полипептид, вовлеченный в биосинтез крахмала», обозначает любой полипептид, понижающая регуляция которого в клетке растения до уровней ниже нормальных (дикий тип), приводит к уменьшению уровня синтеза крахмала и уменьшению уровней крахмала. Это уменьшает переход углерода из сахаров в крахмал. Примером такого полипептида является АГФаза.
В настоящем описании термин «фосфорилаза АДФ-глюкозы» или «АГФаза» обозначает фермент, который регулирует биосинтез крахмала, катализируя перевод глюкозы-1-фосфата и АТФ в АДФ-глюкозу, которая служит строительным блоком для полимеров крахмала. Активная форма фермента АГФазы состоит из 2 больших и 2 малых субъединиц.
Фермент АГФаза в растениях существует, главным образом, в форме тетрамера, который состоит из 2 больших и 2 малых субъединиц. Хотя каталитическая и регуляторная роли этих субъединиц отличаются в зависимости от вида (Kuhn et al., 2009), в растениях малая субъединица проявляет, главным образом, каталитическую активность. Молекулярная масса малой субъединицы составляет приблизительно 50-55 кДа. Последовательности иллюстративных полипептидов малой субъединицы АГФазы представлены в настоящем описании как SEQ ID NO:254-257 (АГФаза Sorghum и Zea mays, No доступа XM_002462095.1 и XM_008666513.1) (Sanjaya et al. 2011, Zale et al. 2016). Молекулярная масса большой субъединицы составляет приблизительно 55-60 кДа. Фермент растения интенсивно активируется 3-фосфоглицератом (PGA), продуктом фиксации диоксида углерода; в отсутствие PGA фермент проявляет только приблизительно 3% активности. Растительная АГФаза также интенсивно ингибируется неорганическим фосфатом (Pi). Наоборот, бактериальная и водорослевая АГФазы существуют в виде гомотетрамеров размером 50 кДа. Водорослевый фермент, подобно его растительному эквиваленту, активируется PGA и ингибируется Pi, тогда как бактериальный фермент активируется фруктозо-1,6-бифосфатом (FBP) и ингибируется АМФ и Pi.
TAG-липазы и бета-окисление
В настоящем описании термин «полипептид, вовлеченный в деградацию липида и/или уменьшающий содержание липида», обозначает любой полипептид, катаболизирующий липид, понижающая регуляция которого в клетке растения до уровней ниже нормальных (дикий тип) приводит к увеличению уровня масла, например, жирных кислот и/или TAG, в клетке трансгенного растения или его части, такой как вегетативная часть, клубень, свекла или семя. Примеры таких полипептидов включают, но без ограничения, липазы или липазу, такую как полипептид CGi58 (подобный сравнительному идентификатору гена-58), сахарозависимую триацилглицерин-липазу 1 (SDP1) (см., например, Kelly et al., 2011) и липазу, описанную в WO 2009/027335.
В настоящем описании термин «TAG-липаза» (EC.3.1.1.3) обозначает белок, который гидролизует TAG до одной или более жирных кислот и любого из DAG, MAG или глицерина. Таким образом, термин «активность TAG-липазы» обозначает гидролиз TAG до глицерина и жирных кислот.
В настоящем описании термин «CGi58» обозначает растворимую ацил-CoA-зависимую ацилтрансферазу лизофосфатидиновой кислоты, кодируемую геном At4g24160 в Arabidopsis thaliana и его гомологами в других растениях, и «Ict1p» в дрожжах и его гомологами. Растительный ген, например, локус гена Arabidopsis At4g24160, экспрессируется как две альтернативные копии: более длинная полноразмерная изоформа (At4g24160.1) и более короткая изоформа (At4g24160.2), в которой отсутствует часть 3' конца (см. James et al., 2010; Ghosh et al., 2009; US 201000221400). Обе иРНК кодируют белок, гомологичный человеческому белку CGI-58 и другим ортологичным членам данного семейства α/β-гидролазы (ABHD). В одном варианте осуществления, белок CGI58 (At4g24160) содержит три мотива, которые являются консервативными в видах растений: мотив липазы GXSXG (SEQ ID NO: 127), мотив ацилтрансферазы HX(4)D (SEQ ID NO: 128), и VX(3)HGF, вероятный мотив связывания с липидом (SEQ ID NO: 129). Белок CGI-58 человека обладает активностью ацилтрансферазы лизофосфатидиновой кислоты (LPAAT), но не активностью липазы. И наоборот, белки растения и дрожжей содержат канонический мотив последовательности липазы GXSXG (SEQ ID NO: 127), отсутствующий в белках позвоночных (человек, мыши и рыбки данио) и обладающий липазной и фосфолипазной активностью (Ghosh et al., 2009). Хотя белки CGI58 растения и дрожжей, похоже, обладают обнаружимыми уровнями активности TAG-липазы и фосфолипазы А, в дополнение к активности LPAAT, белок человека не проявляет такой активности.
Повреждение гомологичного гена CGI-58 в Arabidopsis thaliana приводит к накоплению капелек нейтрального липида в зрелых листьях. Масс-спектроскопический анализ капелек липида, выделенных от мутантов с утратой функции cgi-58,показал, что они содержат триацилглицерины со специфическими жирными кислотами, обычными для листьев. Листья зрелых растений cgi-58 демонстрируют заметное увеличение абсолютных уровней триацилглицеринов, более чем в 10 раза превышающих уровни в растениях дикого типа. Уровни липида в запасающих масло семенах cgi-58 оставались неизменными, и, в отличие от мутаций с β-окислением, семена cgi-58 прорастали и росли нормально, не требуя спасения с помощью сахарозы (James et al., 2010).
Примеры нуклеотидов, кодирующих полипептиды CGi58, включают нуклеотиды из Arabidopsis thaliana (NM_118548.1 кодирующий NP_194147.2; SEQ ID NO:130), Brachypodium distachyon (XP_003578450.1; SEQ ID NO:131), Glycine max (XM_003523590.1 кодирующий XP_003523638.1; SEQ ID NO:132), Zea mays (NM_001155541.1 кодирующий NP_001149013.1; SEQ ID NO:133), Sorghum bicolor (XM_002460493.1 кодирующий XP_002460538.1; SEQ ID NO:134), Ricinus communis (XM_002510439.1 кодирующий XP_002510485.1; SEQ ID NO:135), Medicago truncatula (XM_003603685.1 кодирующий XP_003603733.1; SEQ ID NO:136), и Oryza sativa (кодирующий EAZ09782.1).
В одном варианте осуществления, генетическая модификация по изобретению осуществляет понижающую регуляцию продукции CGi58, где CGi58 кодирован одним или несколькими из следующего:
i) нуклеотидов, содержащих последовательность, приведенную в любой из SEQ ID NO: 130-136,
ii) нуклеотидов, содержащих последовательность, которая по меньшей мере на 30% идентична любой одной или более из SEQ ID NO: 130-136, и
iii) полинуклеотида, который гибридизуется с одним или обоими из i) или ii) в строгих условиях.
Другие липазы, обладающие активностью липазы в отношении TAG, включают сахарозависимую триацилглицерин-липазу 1 (SDP1, см., например, Eastmond et al., 2006; Kelly et al., 2011) и SDP1-подобные полипептиды, обнаруженные в растительных видах, а также клетках дрожжей (полипептид TGL4) и животных, которые вовлечены в расщепление запасенных TAG. По-видимому, SDP1 и SDP1-подобные полипептиды ответственны за начало расщепления TAG в семенах после прорастания (Eastmond et al., 2006). Растения с мутантным SDP1, в отсутствие экзогенных WRI1 и DGAT1, демонстрируют повышенные уровни PUFA в TAG. В настоящем описании «полипептиды SDP1» включают полипептиды SDP1, SDP1-подобные полипептиды и их гомологи в растительных видах. Размер SDP1 и SDP1-подобных полипептидов в растениях составляет 800-910 остатков аминокислот, где они содержат пататин-подобный домен ацилгидролазы, который может связываться с поверхностями масляных телец и предпочтительно, гидролизовать TAG относительно DAG или MAG. Считается, что SDP1 проявляет предпочтение к гидролизу ацильной группы в положении sn-2 TAG. Arabidopsis содержит по меньшей мере три гена, кодирующих SDP1 липазы, а именно SDP1 (номер доступа NP_196024, нуклеотидная последовательность SEQ ID NO: 163 и гомологи в других видах), SDP1 (номер доступа NM_202720 и гомологи в других видах, Kelly et al., 2011) и ATGLL (At1g33270) (Eastmond et al., 2006). Особый интерес с точки зрения снижения активности гена представляют гены SDP1, которые экспрессируются в вегетативных тканях растений, например, в листьях, стеблях и корнях. Таким образом, уровни неполярных липидов в вегетативных частях растения могут быть повышены посредством снижения активности полипептидов SDP1 в частях растения, например, с помощью мутации эндогенного гена, кодирующего полипептид SDP1, или введением экзогенного гена, который кодирует осуществляющую сайленсинг молекулу РНК, которая уменьшает экспрессию эндогенного гена SDP1. Такое снижение приносит конкретную пользу на урожаях клубней, таких как сахарная свекла и картофель, и в «высокосахарозных» растениях, таких как сахарное сорго, сахарный тростник и сахарная свекла.
Гены, кодирующие гомологи SDP1 (в том числе SDP1-подобные гомологи) в выбираемых видах растений, можно легко идентифицировать по гомологии с известными последовательностями гена SDP1. Известные нуклеотидные или аминокислотные последовательности SDP1 включают номера доступа: в Brassica napus, GN078290 (SEQ ID NO:164), GN078281, GN078283; Capsella rubella, XP_006287072; Theobroma cacao, XP_007028574.1; Populus trichocarpa, XP_002308909 (SEQ ID NO:166); Prunus persica, XP_007203312; Prunus mume, XP_008240737; Malus domestica, XP_008373034; Ricinus communis, XP_002530081; Medicago truncatula, XP_003591425 (SEQ ID NO:167); Solanum lycopersicum, XP_004249208; Phaseolus vulgaris, XP_007162133; Glycine max, XP_003554141 (SEQ ID NO:168); Solanum tuberosum, XP_006351284; Glycine max, XP_003521151; Cicer arietinum, XP_004493431; Cucumis sativus, XP_004142709; Cucumis melo, XP_008457586; Jatropha curcas, KDP26217; Vitis vinifera, CBI30074; Oryza sativa, Japonica Group BAB61223; Oryza sativa, Indica Group EAY75912; Oryza sativa, Japonica Group NP_001044325; Sorghum bicolor, XP_002458531 (SEQ ID NO:169); Brachypodium distachyon, XP_003567139 (SEQ ID NO:165); Zea mays, AFW85009; Hordeum vulgare, BAK03290 (SEQ ID NO:172); Aegilops tauschii, EMT32802; Sorghum bicolor, XP_002463665; Zea mays, NP_001168677 (SEQ ID NO:170); Hordeum vulgare, BAK01155; Aegilops tauschii, EMT02623; Triticum urartu, EMS67257; Physcomitrella patens, XP_001758169 (SEQ ID NO:171). Предпочтительные последовательности SDP1 для применения в генетических конструкциях с целью понижающей регуляции экспрессии эндогенных генов, происходят из кДНК, соответствующих генам, которые экспрессируются в наибольшей степени в клетках растений, вегетативных частях растения или семенах, в зависимости от того, что подлежит модификации. Нуклеотидные последовательности, которые являются в высокой степени консервативными между кДНК, соответствующими всем генам SDP1 в виде растений, являются предпочтительными, если желательно уменьшить активность всех членов генного семейства в данном виде.
В одном варианте осуществления, генетическая модификация по изобретению осуществляет понижающую регуляцию продукции SDP1, где SDP1 кодирован одним или несколькими из следующего:
i) нуклеотидов, последовательность которых приведена в любой из SEQ ID NO: 163-174,
ii) нуклеотидов, последовательность которых по меньшей мере на 30% идентична любой одной или более последовательностям, приведенным как SEQ ID NO: 163-174, и
iii) последовательности нуклеотидов, которая гибридизуется с одним или обоими из i) или ii) в строгих условиях.
Как проиллюстрировано в Примерах, снижение экспрессии и/или активности TAG-липазы SDP1 в листьях растения значительно повышало содержание TAG, как с точки зрения количества TAG, которые аккумулируются, так и более раннего наступления периода аккумуляции в ходе развития растения, в контексте коэкспрессии фактора транскрипции WRI1 и Ацилтрансферазы жирных кислот. В частности, увеличение наблюдалось в растениях до цветения, и составляло до приблизительно 70% масс. (% сухой массы) на момент начала старения. Повышение было относительным по сравнению с уровнями TAG, наблюдаемыми в соответствующих листьях растений, трансформированных экзогенными полинуклеотидами, кодирующими WRI1 и ацилтрансферазу жирных ацилов, но без модификации, которая уменьшает экспрессию и/или активность SDP1.
Кроме того, снижение экспрессии других генов катаболизма TAG в частях растения может повысить содержание TAG, например, генов ACX, кодирующих ацил-CoA-оксидазы, таких как гены Acx1 (At4g16760 и гомологи в других видах растений) или Acx2 (At5g65110 и гомологи в других видах растений). Другим полипептид, вовлеченный в катаболизме липидов, является PXA1, который представляет собой пероксисомальный транспортер АТФ-связывающей кассеты, необходимый для импорта жирных кислот для β-окисления (Zolman et al., 2001).
Экспорт жирных кислот из пластид
В настоящем описании термин «полипептид, который увеличивает экспорт жирных кислот из пластид клетки» обозначает любой полипептид, который способствует переносу жирных кислот из внутреннего пространства пластид клеток растений в растении или его части за пределы пластиды, т.е. в любую другую часть клетки, такую как эндоплазматический ретикулум (ER). Примеры таких полипептидов включают, но без ограничения, тиоэстеразу C16 или C18 жирных кислот, такую как полипептид FATA или полипептид FATB, тиоэстеразу C8-C14 жирных кислот (которая дополнительно является полипептидом FATB), транспортер жирных кислот, такой как полипептид ABCA9 или длинноцепочечная ацил-CoA-синтетаза (LACS).
В настоящем описании термина «тиоэстераза жирных кислот» или «FAT», или «ацил-ACP-тиоэстераза» обозначает фермент, катализирующий гидролиз тиоэфирной связи между ацильным фрагментом и ацилпереносящим белком (ACP) в ацил-ACP и высвобождение свободной жирной кислоты. Такие ферменты обычно функционируют в пластидах организма, синтезирующего жирные кислоты de novo. В настоящем описании термин «тиоэстераза C16 или C18 жирной кислоты» обозначает фермент, катализирующий гидролиз тиоэфирной связи между C16 и/или C18 ацильным фрагментом и ACP в ацил-ACP и высвобождение свободной C16 или C18 жирной кислоты в пластиде. Далее свободная жирная кислота переэтерифицируется до CoA в оболочке пластиды, и в таком виде она транспортируется из пластиды. Субстратная специфичность фермента тиоэстеразы жирных кислот (FAT) в пластиде принимает участие в определении спектра длины цепи и степени насыщенности жирных кислот, экспортируемых из пластиды. Ферменты FAT можно классифицировать на два класса, на основании их субстратной специфичности и нуклеотидных последовательностей, FATA и FATB (EC 3.1.2.14) (Jones et al., 1995). Полипептиды FATA отдают предпочтение олеоил-ACP в качестве субстрата, в то время как полипептиды FATB демонстрируют более высокую активность в отношении насыщенных ацил-ACP с различной длиной цепи, например, воздействуя на пальмитоил-ACP с образованием свободной пальмитиновой кислоты. Примеры полипептидов FATA, которые можно использовать по изобретению, включают, но без ограничения, полипептиды из Arabidopsis thaliana (NP_189147), Arachis hypogaea (GU324446), Helianthus annuus (AAL79361), Carthamus tinctorius (AAA33020), Morus notabilis (XP_010104178.1), Brassica napus (CDX77369,1), Ricinus communis (XP_002532744.1) и Camelina sativa (AFQ60946.1). Примеры полипептидов FATB, пригодных в соответствии с изобретением, включают, но без ограничения, полипептиды из Zea mays (AIL28766), Brassica napus (ABH11710), Helianthus annuus (AAX19387), Arabidopsis thaliana (AEE28300), Umbellularia californica (AAC49001), Arachis hypogaea (AFR54500), Ricinus communis (EEF47013) и Brachypodium sylvaticum (ABL85052.1).
В настоящем описании термин «транспортер жирных кислот» относится к полипептиду, присутствующему в мембране пластиды, который принимает активное участие в переносе жирных кислот из пластиды за ее пределы. Примеры полипептидов ABCA9 (ABC транспортер A член семейства 9), пригодных в соответствии с изобретением, включают, но без ограничения, полипептиды из Arabidopsis thaliana (Q9FLT5), Capsella rubella (XP_006279962.1), Arabis alpine (KFK27923.1), Camelina sativa (XP_010457652.1), Brassica napus (CDY23040.1) и Brassica rapa (XP_009136512.1).
В настоящем описании термин «ацил-CoA-синтетаза» или «ACS» (EC 6.2.1.3) обозначает полипептид, который входит в состав семейства лигазы, катализирующего образование жирного ацил-CoA в ходе двухстадийного процесса, проходящего через промежуточное аденилированное соединение, с использованием неэтерифицированной жирной кислоты, CoA и АТФ в качестве субстратов, с образованием эфира ацил-CoA, АМФ и пирофосфата в качестве продуктов. В настоящем описании термин «длинноцепочечная ацил-CoA-синтетаза» (ДACS) обозначает ACS, которая проявляет активность по меньшей мере в отношении субстрата C18 свободной жирной кислоты, хотя она может обладать более широкой активностью в отношении любой из свободных C14-C20 жирных кислот. Эндогенные пластидные ферменты LACS локализуются во внешней мембране пластиды и функционируют вместе с тиоэстеразой жирной кислоты с целью экспорта жирных кислот из пластиды (Schnurr et al., 2002). В Arabidopsis присутствует по меньшей мере девять идентифицированных генов LACS (Shockey et al., 2002). Предпочтительными полипептидами LACS являются принадлежащие к подклассу LACS9, которые в Arabidopsis являются основными пластидными LACS. Примеры полипептидов LACS, пригодных в соответствии с изобретением, включают, но без ограничения, полипептиды из Arabidopsis thaliana (Q9CAP8), Camelina sativa (XP_010416710.1), Capsella rubella (XP_006301059.1), Brassica napus (CDX79212.1), Brassica rapa (XP_009104618.1), Gossypium raimondii (XP_012450538.1) и Vitis Vinifera (XP_002285853.1). Специалист в данной области может легко идентифицировать гомологи вышеупомянутых полипептидов в других видах.
Полипептиды, вовлеченные в продукцию диацилглицерина (DAG)
Уровни неполярных липидов, например, в вегетативных частях растения могут быть дополнительно повышены посредством снижения активности полипептидов, принимающих участие в продукции диацилглицерина (DAG) в пластиде в частях растения, например, мутацией эндогенного гена, кодирующего такой полипептид, или введением экзогенного гена, который кодирует осуществляющую сайленсинг молекулу РНК, уменьшающего экспрессию гена-мишени, вовлеченного в продукцию диацилглицерина (DAG) в пластиде.
В настоящем описании термин «полипептид, вовлеченный в продукцию диацилглицерина (DAG) в пластиде» обозначает любой полипептид в пластиде из клетки растения в растении или его части, который непосредственно вовлечен в синтез диацилглицерина. Примеры таких полипептидов включают, но без ограничения, пластидную GPAT, пластидную LPAAT или пластидную PAP.
GPAT описаны в другом месте настоящего описания. Примеры пластидных полипептидов GPAT, которые могут служить мишенью для понижающей регуляции по изобретению, включают, но без ограничения, полипептиды из Arabidopsis thaliana (BAA00575), Capsella rubella (XP_006306544.1), Camelina sativa (010499766.1), Brassica napus (CDY43010.1), Brassica rapa (XP_009145198.1), Helianthus annuus (ADV16382.1) и Citrus unshiu (BAB79529.1). Специалист в данной области может легко идентифицировать гомологи в других видах.
LPAAT описаны в другом месте настоящего описания. Как понятно специалисту в данной области, пластидные LPAAT, которые служат мишенью для понижающей регуляции с целью снижения синтеза DAG в пластиде, не являются эндогенными LPAAT, которые функционируют за пределами пластиды, например, в ER, например, пригодные для продукции TAG, содержащих среднецепочечные жирные кислоты. Примеры пластидных полипептидов LPAAT, которые могут служить мишенью для понижающей регуляции согласно изобретению, включают, но без ограничения, полипептиды Brassica napus (ABQ42862), Brassica rapa (XP_009137939.1), Arabidopsis thaliana (NP_194787.2), Camelina sativa (XP_010432969.1), Glycine max (XP_006592638.1) и Solanum tuberosum (XP_006343651.1). Специалист в данной области может легко идентифицировать гомологи вышеупомянутых полипептидов в других видах.
В настоящем описании термин «фосфатаза фосфатидиновой кислоты (PAP) (EC 3.1.3.4)» обозначает белок, осуществляющий гидролиз фосфатной группы на 3-sn-фосфатидате с образованием 1,2-диацил-sn-глицерина (DAG) и фосфата. Примеры пластидных полипептидов PAP, которые могут служить мишенью для понижающей регуляции по изобретению, включают, но без ограничения, полипептиды из Arabidopsis thaliana (Q6NLA5), Capsella rubella (XP_006288605.1), Camelina sativa (XP_010452170.1), Brassica napus (CDY10405.1), Brassica rapa (XP_009122733.1), Glycine max (XP_003542504.1) и Solanum tuberosum (XP_006361792.1). Специалист в данной области может легко идентифицировать гомологи вышеупомянутых полипептидов в других видах.
Уровни TAG, например, в вегетативных частях растений, можно также увеличивать посредством увеличения активности полипептидов, вовлеченных в продукцию диацилглицерина (DAG) в ER в частях растения. DAG также образуется в растениях и частях растений по изобретению посредством высвобождения группы DAG из PC и может быть использован для синтеза TAG. Этот DAG обозначен в настоящем описании как «происходящий из PC DAG». Высвобождение может происходить посредством одного или нескольких из ферментов фосфатидилхолин:диацилглицерин-холин-фосфотрансферазы (PDCT), обратной реакции холин:диацилглицерин-холинфосфо-трансферазы (CPT) или фосфолипазы C (PLC), или фосфолипазы D (PLD). Эти ферменты приводят к продукции DAG в ER, а не в пластиде. Как используют в настоящем описании, термин «фосфатидилхолин:диацилглицерин-холинфосфотрансфераза» или «PDCT» (EC 2.7.8.2) обозначает холинфосфотрансферазу, которая осуществляет перенос головной группы фосфохолина из фосфолипида, как правило, PC, для получения DAG, или обратную реакцию для получения PC из DAG. PDCT может, таким образом, осуществлять взаимопревращение PC и DAG (Lu et al., 2009; Hu et al., 2012). Таким образом, двумя субстратами прямой реакции являются цитидинмонофосфат (ЦМФ) и фосфатидилхолин, и двумя продуктами являются ЦДФ-холин и DAG. PDCT принадлежит к семейству белков, родственных фосфатазе фосфатидных кислот, и как правило, имеет домены фосфатазы/фосфотрансферазы липидов (LPT). В Arabidopsis thaliana, PDCT кодирован генами ROD1 (At3g15820) и ROD2 (At3g15830) genes (Lu et al., 2009). Гомологичные гены легко идентифицированы в других видав растений (Guan et al., 2015). Последовательности иллюстративных кодирующих областей и полипептидов PDCT представлены в настоящем описании как SEQ ID NO:262-265 (PDCT Sorghum и Zea mays, номера доступа XM_002437214 и EU973573.1), хотя можно использовать любой кодирующий PDCT ген. Иллюстративные полипептиды PDCT имеют аминокислотные последовательности, представленные номерами доступа. NP_566527.1 (Arabidopsis), XP_010487422.1 (Camelina sativa), XP_003531718.1 (Glycine max), XP_013695400,1 (Brassica napus), XP_012073167.1 (Jatropha curcas), XP_002517643.1 (Ricinus communis) XP_013587626.1 (Brassica oleracea), XP_016725741.1 и XP_016725742.1 (Gossypium hirsutum), AQK82308.1, NP_001145186.1 (Zea mays), и XP_021306179.1 (Sorghum bicolor). Гомологи и природные варианты из этих или других видов растений, грибов или водорослей, можно легко идентифицировать и использовать по настоящему изобретению. В одном варианте осуществления, гомолог или вариант является по меньшей мере на 95% идентичным, предпочтительно по меньшей мере 99% идентичным, аминокислотной последовательности из перечисленных номеров доступа. В одном варианте осуществления, PDCT является отличным от PDCT A. thaliana (Lu et al., 2009). Увеличенная экспрессия PDCT, который может являться экзогенным или эндогенным для клетки или растения по изобретению, и который, предпочтительно, экспрессирован с экзогенного полинуклеотида, увеличивает поток этерифицированных ацильных групп от PC к DAG и таким образом, увеличивает TTQ в общем содержании жирных кислот и уровень TAG в вегетативных частях растений или клеток по изобретению. Альтернативно, уменьшение уровня активности PDCT в клетке или растении посредством мутации в гене или посредством осуществляющей сайленсинг молекулы РНК, уменьшает получение PC из DAG, обратную реакцию PDCT, и позволяет использование большего количества de novo DAG для синтеза TAG.
Фермент PDCT обеспечивает перенос 18:1 от DAG к PC для десатурации, а также обратный перенос полиненасыщенных жирных кислот 18:2 и 18:3 из PC к DAG, который можно использовать для синтеза TAG. Эксперименты с мечением жирных кислот показывают, что de novo DAG и происходящий из PC DAG представлены как два отдельных пула DAG (Bates 2016), возможно, сохраняемые пространственно разделенными в ER. Некоторые сообщения позволяют предполагать, что происходящий из PC DAG может являться преобладающей формой DAG, используемой в синтезе TAG (Bates and Browse, 2011).
Как используют в настоящем описании, термин «ЦДФ-холин:диацилглицерин-холинфосфо-трансфераза» или «CPT» (EC 2/7/8/2), обозначает фермент, катализирующий перенос группы холина от ЦДФ-холина к DAG, образуя PC и ЦМФ. Эта прсилосная ямая реакция приводит к чистому синтезу PC из DAG. CPT катализирует также обратную реакцию, образующую DAG из PC, возможно, обеспечивая равновесие уровней DAG и PC в клетке. Arabidopsis содержит два гена (AtAAPT1 и AtAAPT2, номера доступа AAC61768.1 и AAC61769.1), кодирующие ферменты CPT. Мутации в любом гене влияют на гомеостаз мембраны, и двойной мутант является летальным (Liu et al., 2015). Иллюстративные ферменты CPT имеют аминокислотные последовательности, представленные следующими номерами доступа: XP_010495346.1 и XP_019084815.1 (Camelina sativa); XP_013731103,1 и XP_013720632.1 (Brassica napus); XP_013585950.1 (Brassica oleracea); XP_015572083.1 (Ricinus communis); XP_016679754,1 (Gossypium hirsutum); XP_010687532.1 (Beta vulgaris); XP_012081980.1 (Jatropha curcas); XP_011070871.1 (Sesamum indicum); NP_001151915.1 и XP_008649199.1 (Zea mays); XP_002451408.1 и XP_021305900.1 (Sorghum bicolor). Гомологи и природные варианты из этих или других видов растений, грибов или водорослей, можно легко идентифицировать и использовать по настоящему изобретению. В одном варианте осуществления, гомолог или вариант является по меньшей мере на 95% идентичным, предпочтительно по меньшей мере 99% идентичным, аминокислотной последовательности из перечисленных номеров доступа.
Фосфолипазы представляют собой ферменты, гидролизующие фосфолипиды до таких продуктов, как фосфатидная кислота (PA), DAG, свободные жирные кислоты (FFA) или лизофосфолипиды (LPL), в зависимости от класса фосфолипаз. Как используют в настоящем описании, термин «фосфолипаза C» или «PLC» обозначает фермент, катализирующий расщепленте фосфолипида с формированием DAG и фосфорилированной головной группы, где расщепление происходит по сложноэфирной связи между фосфатной группой и глицериновым остовом фосфолипида. Фосфорилированная головная группа представляет собой фосфохолин, когда фосфолипид представляет собой PC. PLC отличаются от ферментов фосфолипазы D, которые отщепляют головную группу фосфолипидов с образованием фосфатидной кислоты (PA), а не DAG в качестве продуекта, и от фосфолипазы A1 и фосфолипазы A2, образующих FFA из фосфолипидоы. PLC представляют собой ассоциированные с мембраной ферменты, обнаруженные в широком множестве растений, животных и прокариот. PLC можно разделять на три класса, специфические для фосфатидилинозитола фосфолипазы C (PI-PLC), специфические для фосфатидилхолина фосфолипазы C (PC-PLC) и PLC, гидролизующие заякоренные в гликозилфосфатидилинозитоле (GPI) белки (GPI-PLC), в соотвтествии с диапазоном их субстратной специфичности (Pokotylo et al., 2013, Hong et al., 2016). Мультидоменные PI-PLC животных представляют собой активированные G-белком ферменты, которые регулируют уровни кальция и протеинкиназу C, и таким образом, являются ключевыми компонентаи регуляторных систем клеточного роста и развития. PI-PLC являются менее предпочтительными по настоящему изобретению. PC-PLC, в растениях также известные как неспецифические PLC (NPC), имеют более широкие диапазоны субстратов и, как правило, являются наиболее активными по отношению PC, и, таким образом, являются предпочтительными по настоящему изобретению. PC-PLC идентифицированы в бактериях (Titball 1993), грибах (Morelle et al., 2005) и растениях (Hong et al., 2016), где ферменты из растений являются более предпочтительными. Шесть генов PC-PLC идентифицированы в Arabidopsis (Wang, 2001; Nakamura et al., 2005), девять генов в сое (Huang et al., 2010), пять генов в рисе (Singh et al., 2013) и множество копий в разнообразных видах растений. PC-PLC растений классифицированы на несколько подгрупп, а именно, подгруппы NPC1, NPC2, NPC3-5 и NPC6 (Pokotylo et al., 2013), на основании гомологии с аминокислотными последовательностями Arabidopsis, имеющие также различные (но перекрывающиеся) субстратные специфичности и распределения в тканях. Например, для NPC4 Arabidopsis показана активность по отношению к PC и PE, небольшая активность по отношению к PS, НО НЕ К PA и PIP2. NPC5 являлся способным расщеплять PC и PE, в то время как для NPC3 показана активность фосфатазы лизофосфатидной кислоты (LPA), приводящая к продукции MAG, так же как к расщеплению PC. NPC4 экспрссировался в зрелых листьях, и NPC6 экспрессировался в большинстве тканей. PLC Arabidopsis имеют между 510 и 540 аминокислотных остатков. В растениях, PLC вовлечены в ремоделирование липидов и в ответы растений на недостаточность фосфата и осмотический, солевой и тепловой стрессы, среди других функций. Примеры PLC включают идентифицированные PLC, с аминокислотными последовательностями под следующими номерами доступа, из Arabidopsis: NPC1, NP_172203.2; NPC2, NP_180255.1; NPC4, NP_566206.1; NPC5, NP_566207.1; NPC6, NP_190430.2; из Camelina sativa, NPC1, XP_010457889.1; NPC2, XP_010473071.1; NPC4, XP_010463802.1; NPC5, XP_010485694.1; NPC6, XP_010426358.1; из Brassica napus NPC1, XP_013687149.1; NPC2, XP_013744020.1; NPC4, XP_013682889.1; NPC6, XP_002511167.1; из Ricinus communis NPC1, XP_002525632.1; NPC4, XP_002524007.1; NPC6, XP_002511167.1; из Gossypium hirsutum NPC1, XP_016715492.1; NPC2, XP_016745351.1; NPC4, XP_016697678.1; NPC6, XP_016734150.1; из Beta vulgaris NPC1, XP_010685575.1; NPC2, XP_010673757.1; NPC4, XP_010691936.1; гомолог NPC1 из Zea mays, NP_001170209.1; из Oryza sativa NPC1, XP_015631592.1; из Glycine max NPC1, XP_003551286.1; NPC2, XP_003556783.1; NPC4, XP_003521010.1; NPC6, XP_003523153.1; NPC6, XP_003526950.1; из Jatropha curcas NPC2, XP_012065897.1; NPC4, XP_012070293.1; NPC6, XP_012090681.1; из Solanum tuberosum NPC2, XP_006362756,1; из Elaeis guineensis NPC2, XP_010938556.1; NPC4, XP_010919939,1; из Brachypodium distachyon NPC2, XP_003565100.1; из Trifolium subterraneum NPC4, GAU26202.1; NPC6, GAU26767.1; из Medicago truncatula NPC4, XP_013444051.1; и из Helianthus annuus NPC6, XP_021984157.1. Гомологи и природные варианты из этих или других видов растений, грибов или водорослей, можно легко идентифицировать и использовать по настоящему изобретению. В одном варианте осуществления, гомолог или вариант является по меньшей мере на 95% идентичным, предпочтительно по меньшей мере 99% идентичным, аминокислотной последовательности из перечисленных номеров доступа.
Как используют в настоящем описании, термин «фосфолипаза D» или «PLD» обозначает фермент, катализирующий расщепление фосфодиэфирной связи головной группы мембраного фосфолипида с формированием фосфатидной кислоты (PA) и растворимой головной группы, где расщепление происходит на фосфодиэфирной связи, наиболее отдаленной от глицеринового остова фосфолипида. Продукт растворимой головной группы представляет собой холин, когда фосфолипид представляет собой PC. PA затем переводят в DAG под действием PAP. Множество PLD идентифицировано в растениях, животных, грибах и прокариотах. Все анализированные виды растений имеют семейство PLD по меньшей мере из 10 генов PLD (Wang, 2005), например, семейство PLD Arabidopsis имеет 12 идентифицированных генов. PLD классифицированы на подгруппы α, β, γ, δ, ε и ζ, и на основании последовательности и ферментативных свойств (Hong et al, 2016). PLD имеют либо домен C2 из приблизительно 130 аминокислот, вовлеченный в связывание иона кальция и фосфолипида или гомологичный плекстрину (PH) домен и гомологичный phox (PX) домен. Все анализированные эукариотические PLD содержат два дуплицированных каталитических мотива HKD, которые разделены более чем 300 аминокислотами в PLD Arabidopsis, но которые взаимодействуют друг с другом с формированием активного участка (Hong et al, 2013). Иллюстративные PLD включают PLD из Arabidopsis: номера доступа. AAL06337.1, CAJ58441.1; из Camelina sativa, XP_010465702.1, XP_010430169.1; из Gossypium hirsutum, XP_016724300.1; из Ricinus communis, XP_015573380.1; из Solanum lycopersicum, XP_004229274.1; из Jatropha curcas, XP_012083994.1; из Glycine max, XP_003534832.1, NP_001275522,1; из Elaeis guineensis, XP_010921600.1; из Brassica rapa, XP_009146059.1; из Beta vulgaris, XP_010693582.1 и XP_016713715.1; Medicago truncatula XP_003591178.2; и из Brassica napus, XP_013677646.1. Гомологи и природные варианты из этих или других видов растений, грибов или водорослей, можно легко идентифицировать и использовать по настоящему изобретению. В одном варианте осуществления, гомолог или вариант является по меньшей мере на 95% идентичным, предпочтительно по меньшей мере 99% идентичным, аминокислотной последовательности из перечисленных номеров доступа.
Импорт жирных кислот в пластиды
Уровни неполярных липидов в вегетативных частях растения могут быть дополнительно увеличены посредством снижения активности полипептидов TGD в частях растения, например, с помощью мутации эндогенного гена, кодирующего полипептид TGD, или введения экзогенного гена, кодирующего осуществляющую сайленсинг молекулу РНК, которая уменьшает экспрессию эндогенного гена TGD. В настоящем описании «полипептид тригалактозилдиацилглицерина» (TGD) представляет собой полипептид, принимающий участие в транспорте липидов к хлоропластам в ER (Xu et al., 2010; Fan et al., 2015)) и принимающий участие в формировании белкового комплекса, осуществляющего функцию пермеазы для липидов. Четыре таких полипептида известны, как формирующие или связанные с TGD-пермеазой, а именно TGD-1 (номер доступа At1g19800 и гомологи в других видах), TGD-2 (номер доступа At2g20320 и гомологи в других видах), TGD-3 (номер доступа NM-105215 и гомологи в других видах) и TGD-4 (At3g06960 и гомологи в других видах) (US 20120237949). TGD5 также вовлечена в транспорт липидов к хлоропластам в ER, и понижающая регуляция TGD5 ассоциирована с увеличенной продукцией масла (US2015/337017; Fan et al., 2015). Последовательности иллюстративных полипептидов TGD5 представлены в настоящем описании как SEQ ID NO:250-253 (TGD5 Sorghum and Zea mays, номера доступа XM_002442154 and EU972796.1). Считается, что полипептиды TGD-1, -2 и -3 являются компонентами транспортера АТФ-связывающей кассеты (АСК), связанного с внутренней мембраной оболочки хлоропласта. Полипептиды TGD-2 и TGD-4 связываются с фосфатидиновой кислотой, тогда как полипептид TGD-3 действует, как АТФаза в строме хлоропласта. В настоящем описании «эндогенный ген TGD» представляет собой ген, кодирующий полипептид TGD в растении. Мутации в гене TGD-1 A. thaliana вызывали накопление триацилглицеринов, олигогалактолипидов и фосфатидиновой кислоты (PA) (Xu et al., 2005). Мутации в генах TGD или генах SDP1, или, в действительности, в любом желательном гене в растении, можно вводить в сайт-специфической форме с помощью искусственной цинк-пальцевой нуклеазы (ZFN), эффектора TAL (TALEN) или технологии CRISPR (с использованием нуклеазы типа Cas9), как известно в данной области. Предпочтительными экзогенными генами, кодирующими осуществляющие сайленсинг РНК, являются гены, кодирующие молекулу двухцепочечной РНК, такой как шпилечная РНК или искусственный предшественник микроРНК.
Метаболизм сахарозы
Уровни TAG и/или TTQ в общем содержании жирных кислот в клетках, растениях и частях растений по изобретению, можно также увеличивать посредством модификации метаболизма сахарозы, в частности, в стеблях растений, таких как сахарный тростник, Sorghum и Zea mays. В одном варианте осуществления, этого достигают посредством увеличения экспрессии полипептида метаболизма сахарозы, такого как инвертаза или синтаза сахарозы, или полипептида для транспорта сахарозы, такого как SUS1, SUS4, SUT2, SUT4 или SWEET. Эффектом этих полипептидов является увеличение поставки сахарозы и ее моносахаридных компонентов в цитозоль клеток и/или увеличение транспорта и/или хранения сахарозы в вакуолях клеток, в частности, в клетках стебля. Последовательности примеров этих полипептидов представлены в SEQ ID NO:274-292. Инвертаза, такая как bCIN, INV2 или INV3, действует для перевода сахарозы в гексозы, которые могут подвергаться экспорту из вакуолей в цитоплазму (McKinley et al., 2016). Увеличенная экспрессия SUS1 или SUS4 расщепляет цитозольную сахарозу в гексозах для гликолиза и синтеза жирных кислот de novo, вместо транстпорта сахарозы в вакуоли, например, в клетках паренхимы стебля (McKinley et al., 2016). Увеличенная экспрессия полипептидов транспорта сахаров, таких как экспортер сахарозы тонопластов, например, полипептида SUT2 или SUT4, или SWEET, высвобождает вакуолярную сахарозу для цитозольного гликолиза и увеличивает биосинтез жирных кислот de novo (Bihmidine et al., 2016; Qazi et al., 2012; Schneider et al., 2012; Hedrich et al., 2015; Klemens et al., 2013).
Уровни TAG и/или TTQ в общем содержании жирных кислот в клетках, растениях и частях растений по изобретению также можно увеличивать посредством уменьшения уровня полипептидов TST, таких как TST1 или TST2, в частности, в стеблях растений, таких как сахарный тростник, Sorghum и Zea mays. Уровень полипептида TST можно уменьшать посредством мутации эндогенных генов, кодирующих полипептид, или посредством введения экзогенного полинуклеотида, который кодирует осуществляющую сайленсинг молекулу РНК. Последовательности иллюстративных кДНК и полипептидов TST представлены как SEQ ID NO:266-273.
Ферменты, модифицирующие жирные кислоты
В настоящем описании термин «FAD2» обозначает связанную с мембраной дельта-12 десатуразу жирной кислоты, которая образует кратные связи в олеиновой кислоте (C18:1Δ9)с образованием линолевой кислоты (C18:2Δ9,12).
В настоящем описании термин «эпоксигеназа» или «эпоксигеназа жирной кислоты» обозначает фермент, который вводит эпоксидную группу в жирную кислоту, с получением в результате эпоксижирной кислоты. В предпочтительном варианте осуществления эпоксигруппа вводится при 12-м атоме углерода на цепи жирной кислоты, если эпоксигеназа представляет собой Δ12-эпоксигеназу, особенно цепи жирной кислоты C16 или C18. Эпоксигеназа может быть Δ9-эпоксигеназой, Δ15-эпоксигеназой или оказывать свое действие при другом положении в цепи ацила, как известно в данной области. Эпоксигеназа может принадлежать к классу P450. Предпочтительные эпоксигеназы принадлежат к классу монооксигеназ, как описано в WO98/46762. Многочисленные эпоксигеназы или предполагаемые эпоксигеназы клонированы и известны в данной области. Другие примеры эпоксигеназ включают белки, содержащие последовательность аминокислот, представленную в SEQ ID NO: 21 WO 2009/129582, полипептиды, кодируемые генами Crepis paleastina (CAA76156, Lee et al., 1998), Stokesia laevis (AAR23815) (монооксигеназный тип), Euphorbia lagascae (AAL62063) (тип P450), CYP2J2 человека (эпоксигеназа арахидоновой кислоты, U37143); CYPIA1 человека (эпоксигеназа арахидоновой кислоты, K03191), а также их варианты и/или мутанты.
В настоящем описании термин «гидроксилаза» или «гидроксилаза жирной кислоты» обозначает фермент, который вводит гидроксильную группу в жирную кислоту, с образованием гидроксилированной жирной кислоты. В предпочтительном варианте осуществления гидроксильная группа вводится при 2-м, 12-м и/или 17-м атоме углерода на цепи жирной кислоты C18. Предпочтительно, гидроксильная группа вводится при 12 -м атоме углерода, если гидроксилаза представляет собой Δ12-гидроксилазу. В другом предпочтительном варианте осуществления гидроксильная группа вводится при 15-м атоме углерода на цепи жирной кислоты C16. Дополнительно, гидроксилазы могут обладать активностью фермента, такого как десатураза жирной кислоты. Примеры генов, кодирующих Δ12-гидроксилазы включают полученные из Ricinus communis (AAC9010, van de Loo 1995); Physaria lindheimeri, (ABQ01458, Dauk et al., 2007); Lesquerella fendleri, (AAC32755, Broun et al., 1998); Daucus carota, (AAK30206); гидроксилазы жирных кислот, которые гидроксилируют конец жирных кислот, например: A, thaliana CYP86A1 (P48422, ω-гидроксилаза жирной кислоты); Vicia sativa CYP94A1 (P98188, ω-гидроксилаза жирной кислоты); CYP2E1 мыши (X62595, ω-1 гидроксилаза лауриновой кислоты); CYP4A1 крысы (M57718, ω-гидроксилаза жирной кислоты), а также их варианты и/или мутанты.
В настоящем описании термин «конъюгаза» или «конъюгаза жирной кислоты» обозначает фермент, способный к образованию конъюгационной связи в ацильной цепи жирной кислоты. Примеры конъюгаз включают конъюгазы, кодируемые генами Calendula officinalis (AF343064, Qiu et al., 2001); Vernicia fordii (AAN87574, Dyer et al., 2002); Punica granatum (AY178446, Iwabuchi et al., 2003) и Trichosanthes kirilowii (AY178444, Iwabuchi et al., 2003); а также их варианты и/или мутанты.
В настоящем описании термин «ацетиленаза» или «ацетиленаза жирной кислоты» обозначает фермент, который вводит тройную связь в жирную кислоту, что приводит в результате к образованию ацетиленсодержащей жирной кислоты. В предпочтительном варианте осуществления тройная связь вводится при 2-м, 6-м, 12-м и/или 17-м атоме углерода на цепи жирной кислоты C18. Примеры ацетиленаз включают полученные из Helianthus annuus (AA038032, ABC59684), а также их варианты и/или мутанты.
Примеры таких модифицирующих жирные кислоты генов включают белки в соответствии со следующими номерами доступа, которые сгруппированы по предполагаемой функции, и гомологи из других видов: Δ12-ацетиленазы ABC00769, CAA76158, AAO38036, AAO38032; Δ12 конъюгазы AAG42259, AAG42260, AAN87574; Δ12-десатуразы P46313, ABS18716, AAS57577, AAL61825, AAF04093, AAF04094; Δ12 эпоксигеназы XP_001840127, CAA76156, AAR23815; Δ12-гидроксилазы ACF37070, AAC32755, ABQ01458, AAC49010; и ферменты Δ12 P450, например, AF406732.
Супрессоры сайленсинга
В одном варианте осуществления, трансгенное растение или его часть по изобретению может содержать супрессор сайленсинга.
В настоящем описании «супрессор сайленсинга» увеличивает экспрессию трансгена в растении или его части по изобретению. Например, присутствие супрессора сайленсинга приводит к более высоким уровням полипептида(полипептидов), продуцируемого экзогенным полинуклеотидом(полинуклеотидами) в растении или его части по изобретению, по сравнению с соответствующей клеткой, не содержащей супрессора сайленсинга. В одном варианте осуществления, супрессор сайленсинга предпочтительно, связывается с молекулой дцРНК, размер которой составляет 21 пару оснований, относительно молекулы дцРНК другой длины. Это является признаком по меньшей мере супрессора сайленсинга типа p19, а именно p19 и его функциональных ортологов. В другом варианте осуществления супрессор сайленсинга предпочтительно, связывается с двухцепочечной молекулой РНК с нависающими 5' концами, по сравнению с соответствующей двухцепочечной молекулой РНК с тупыми концами. Это признак супрессора сайленсинга типа V2, а именно V2 и его функциональных ортологов. В одном варианте осуществления, молекула дцРНК или ее процессированный РНК продукт содержит по меньшей мере 19 нуклеотидов подряд, длина которого предпочтительно, составляет 19-24 нуклеотида, с 19-24 парами оснований подряд в случае двухцепочечной молекулы шпилечной РНК или процессированного РНК продукта, более предпочтительно, состоит из 20, 21, 22, 23 или 24 нуклеотидов и предпочтительно, содержит метилированный нуклеотид, по меньшей мере на 95% идентичный последовательности, комплементарной участку РНК-мишени, где, участок РНК-мишени находится i) в пределах 5' нетранслируемого участка РНК-мишени, ii) в пределах 5' половины РНК-мишени, iii) в пределах кодирующей белок открытой рамки считывания РНК-мишени, iv) в пределах 3' половины РНК-мишени или v) в пределах 3' нетранслируемого участка РНК-мишени.
Дополнительные подробности относительно супрессоров сайленсинга хорошо известны из уровня техники и описаны в WO 2013/096992 и WO 2013/096993.
Полинуклеотиды
Термины «полинуклеотид», и «нуклеиновая кислота» используются взаимозаменяемо. Они обозначают полимерную форму нуклеотидов любой длины, дезоксирибонуклеотидов или рибонуклеотидов или их аналогов. Полинуклеотид по изобретению может быть геномного, кДНК, полусинтетического или синтетического происхождения, одноцепочечным или двухцепочечным, и вследствие его происхождения или манипуляций с ним: (1) не связан с полноразмерным или частью полинуклеотида, с которым он связан в природе, (2) связан с другим полинуклеотидом, чем тот, с которым он связан в природе, или (3) не встречается в природе. Ниже приведены неограничивающие примеры полинуклеотидов: кодирующие или не кодирующие участки гена или фрагмента гена, экзоны, интроны, информационная РНК (иРНК), транспортная РНК (тРНК), рибосомальная РНК (рРНК), рибозимы, кДНК, рекомбинантные полинуклеотиды, плазмиды, векторы, выделенная ДНК с любой последовательностью, выделенная РНК с любой последовательностью, химерная ДНК с любой последовательностью, зонды нуклеиновой кислоты и праймеры. Для применения in vitro полинуклеотид может включать нуклеотиды, модифицированные, например, посредством конъюгации с меткой.
В настоящем описании «выделенный полинуклеотид» обозначает полинуклеотид, который отделен от полинуклеотидных последовательностей, с которыми он связан или соединен в природном состоянии, или от неприродного полинуклеотида.
В настоящем описании термин «ген» понимается в самом широком контексте и включает дезоксирибонуклеотидные последовательности, содержащие транскрибированный участок и, при условии трансляции, кодирующая область белка структурного гена и, в том числе, последовательности, смежные с кодирующим участком, на 5' и 3' концах, на расстоянии по меньшей мере приблизительно 2 тысяч пар оснований на любом конце, которые принимают участие в экспрессии гена. В этом отношении, ген содержит контрольные сигналы, например, промоторы, энхансеры, сигналы терминации и/или полиаденилирования, которые в природе связаны с указанным геном, или гетерологичные контрольные сигналы, и в этом случае ген обозначается, как «химерный ген». Последовательности, которые размещены в направлении 5' по отношению к кодирующему белок участку, и которые присутствуют в иРНК, обозначаются как 5' нетранслируемые последовательности. Последовательности, которые размещены в направлении 3' или по ходу транскрипции относительно кодирующего белок участка, и которые присутствуют в иРНК, обозначаются, как 3' нетранслируемые последовательности. Термин «ген» включает кДНК и геномные формы гена. Геномная форма или клон гена содержит кодирующий участок, который может перемежаться не кодирующими последовательностями под названием «интроны», «вставочные последовательности» или «встроенные последовательности». Интроны представляют собой фрагменты гена, которые транскрибированы в ядерную РНК (яРНК). Интроны могут содержать регуляторные элементы, например, энхансеры. Интроны удаляются, или «происходит укорачивание» ядерной или первичной копии; таким образом, интроны отсутствуют в копии иРНК. Ген, содержащий по меньшей мере один интрон, может подвергаться вариабельному сплайсингу с образованием альтернативных иРНК из одного транскрибированного гена и, таким образом, полипептидных вариантов. В природном или химерном гене интроны могут отсутствовать. Функции иРНК в ходе трансляции состоят в определении последовательности или порядка аминокислот в возникающем полипептиде. Термин «ген» включает синтетическую или слитую молекулу, кодирующую полноразмерные белки по изобретению или их части, как описано в настоящем описании, и нуклеотидную последовательность, комплементарную к любому из упомянутого выше.
В настоящем описании «химерная ДНК» обозначает любую молекулу ДНК, которая не обнаружена в природе; также обозначается в настоящем описании как «конструкция ДНК» или «генетическая конструкция». Обычно, химерная ДНК содержит регуляторную и транскрибированную или кодирующую белок последовательности, которые не обнаружены вместе в природе. Соответственно, химерная ДНК может содержать регуляторные последовательности и кодирующие последовательности, которые происходят из различных источников или регуляторных последовательностей, и кодирующие последовательности, происходящие из того же источника, но организованные до некоторой степени отличным образом, чем обнаруженные в природе. Открытая рамка считывания может быть или может не быть связанной с ее природными регуляторными элементами, расположенными против хода транскрипции и по ходу транскрипции. Открытая рамка считывания может быть инкорпорирована, например, в геном растения, в неприродном расположении или в репликоне или векторе, где она не обнаружена в природе, например, бактериальной плазмиде или вирусном векторе. Термин «химерная ДНК» не ограничивается молекулами ДНК, которые являются реплицируемыми в организме-хозяине, но включает ДНК, способную к лигированию в репликон, например, специфическими адаптерными последовательностями.
«Трансген» представляет собой ген, который введен в геном посредством способа трансформации. Термин включает ген в растении-потомке или его части, такой как вегетативная часть, который был введен в геном их родительской клетки. Такие клетки-потомки, и т. п. могут представлять по меньшей мере 3-е или 4-е поколение потомков относительно родительской клетки, которая была первоначально трансформирована, или родительского трансгенного растения (которое в настоящем описании обозначается как растение T0). Потомок может быть получен посредством полового воспроизводства или вегетативно, например, из клубней картофеля или отводков сахарного тростника. Термин «генетически модифицированный», «генетическая модификация» и их варианты представляют более широкий термин, который включает введение гена в клетку посредством трансформации или трансдукции, видоизменение гена в клетке и генную модификацию или модуляцию регулирования гена в клетке или потомке любой модифицированной клетки, как описано выше.
«Геномная область» в настоящем описании обозначает положение в пределах генома, в котором трансген или группа трансгенов (также обозначается в настоящем описании как кластер) вставлены в клетку или ее предка. Такие области содержат только нуклеотиды, введенные в результате вмешательства человека, например, способами, описанными в настоящем описании.
«Рекомбинантный полинуклеотид» по изобретению обозначает молекулу нуклеиновой кислоты, которая сконструирована или модифицирована способами искусственной рекомбинации. Рекомбинантный полинуклеотид может присутствовать в клетке растения или его части в измененном количестве или экспрессироваться с измененной скоростью (например, в случае иРНК), по сравнению с его природным состоянием. В одном варианте осуществления полинуклеотид введен в клетку, которая от природы не содержит полинуклеотида. Обычно экзогенная ДНК используется в качестве шаблона для транскрипции иРНК, которая в дальнейшем транслируется в непрерывную последовательность остатков аминокислоты, кодируя полипептид по изобретению в пределах трансформированной клетки. В другом варианте осуществления полинуклеотид является эндогенным для растения или его части, и его экспрессия модифицирована рекомбинантными способами, например, экзогенная контрольная последовательность введена в направлении относительного целевого эндогенного гена, чтобы предоставить возможность трансформированному растению или его части экспрессировать полипептид, кодируемый геном, или делеция введена в представляющий интерес ген способами ZFN, TALEN или CRISPR.
Рекомбинантный полинуклеотид по изобретению содержит полинуклеотиды, которые не были отделены от других компонентов клеточной или неклеточной системы экспрессии, в которой он присутствует, и полинуклеотиды, образованные в указанных клеточных или неклеточных системах, которые впоследствии очищают по меньшей мере от некоторых других компонентов. Полинуклеотид может быть беспрерывным участком нуклеотидов или может содержать два или более смежных участков нуклеотидов из различных источников (природных и/или синтетических), соединенных таким образом, чтобы сформировать единый полинуклеотид. Обычно, такие химерные полинуклеотиды содержат по меньшей мере открытую рамку считывания, кодирующую полипептид по изобретению, функционально связанную с промотором, пригодным для управления транскрипцией открытой рамки считывания в представляющей интерес клетке.
Применительно к определенным полинуклеотидам, необходимо понимать, что % идентичности, превышающий приведенные выше, будет охватывать предпочтительные варианты осуществления. Таким образом, если это уместно, в свете минимальных значений % идентичности, предпочтительно, полинуклеотид содержит последовательность полинуклеотида, которая по меньшей мере на 60%, более предпочтительно, по меньшей мере на 65%, более предпочтительно, по меньшей мере на 70%, более предпочтительно, по меньшей мере на 75%, более предпочтительно, по меньшей мере на 80%, более предпочтительно, по меньшей мере на 85%, более предпочтительно, по меньшей мере на 90%, более предпочтительно, по меньшей мере на 91%, более предпочтительно, по меньшей мере на 92%, более предпочтительно, по меньшей мере на 93%, более предпочтительно, по меньшей мере на 94%, более предпочтительно, по меньшей мере на 95%, более предпочтительно, по меньшей мере на 96%, более предпочтительно, по меньшей мере на 97%, более предпочтительно, по меньшей мере на 98%, более предпочтительно, по меньшей мере на 99%, более предпочтительно, по меньшей мере на 99,1%, более предпочтительно, по меньшей мере на 99,2%, более предпочтительно, по меньшей мере на 99,3%, более предпочтительно, по меньшей мере на 99,4%, более предпочтительно, по меньшей мере на 99,5%, более предпочтительно, по меньшей мере на 99,6%, более предпочтительно, по меньшей мере на 99,7%, более предпочтительно, по меньшей мере на 99,8%, и даже более предпочтительно, по меньшей мере на 99,9% идентична представленной в соответствующей SEQ ID NO.
Полинуклеотид по настоящему изобретению или пригодный для применения в соответствии с настоящим изобретением может избирательно гибридизоваться, в строгих условиях, с полинуклеотидом, определенным в настоящем описании. В настоящем описании строгие условия представляют собой: (1) применение в ходе гибридизации денатурирующего агента, такого как формамид, например, 50%, об/об формамида, содержащего 0,1%, масс/об телячьего сывороточного альбумина, 0,1% Фиколла, 0,1% поливинилпирролидона, 50 мМ натрий фосфатного буфера, pH 6,5, содержащего 750 мМ NaCl, 75 мМ натрия цитрата, при 42°C; или (2) применение 50%, формамида, 5 x натрия хлорида+натрия цитрата (0,75 M NaCl, 0,075 M натрия цитрата), 50 мМ натрия фосфата (pH 6,8), 0,1% натрия пирофосфата, 5 x раствор Денхардта, обработанная ультразвуком ДНК спермы лосося (50 г/мл), 0,1% натрия лаурилсульфата и 10% декстрана сульфата при 42°C в 0,2 x натрия хлорида+натрия цитрата и 0,1% натрия лаурилсульфата, и/или (3) применение низкой ионной силы и высокой температуры для промывания, например, 0,015 M NaCl/0,0015 M натрия цитрата/0,1% натрия лаурилсульфата при 50°C.
Полинуклеотиды по изобретению могут содержать, по сравнению с природными молекулами, одну или более мутаций, которые представляют собой делеции, вставки или замены нуклеотидных остатков. Полинуклеотиды, которые содержат мутации относительно эталонной последовательности, могут быть природными (т. е., выделенными из природного источника) или синтетическими (например, полученными посредством сайт-направленного мутагенеза или перетасовки ДНК в нуклеиновой кислоте, как описано выше).
Полинуклеотиды для уменьшения уровней экспрессии генов
РНК-интерференция
РНК-интерференция (РНКи) особенно пригодна для специфичного снижения экспрессии гена, что ведет к уменьшению продукции конкретного белка, если ген кодирует белок. Не желая ограничиваться теорией, Waterhouse et al. (1998) предложили модель механизма, по которому двухцепочечная РНК (дцРНК) может использоваться для снижения продукции белка. Данная технология зависит от присутствия молекул дцРНК, которые содержат последовательность, по существу идентичную иРНК целевого гена или его части. Пригодным образом, дцРНК может быть получена с помощью единственного промотора в рекомбинантном векторе или клетке-хозяине, в которых смысловые и антисмысловые последовательности фланкированы посторонней последовательностью, которая позволяет смысловой и антисмысловой последовательностям гибридизоваться с образованием молекулы дцРНК с посторонней последовательностью, образующей структуру петли. Конструирование и получение подходящих молекул дцРНК находится в пределах компетенции специалиста в данной области, особенно с учетом Waterhouse et al. (1998), Smith et al. (2000), WO 99/32619, WO 99/53050, WO 99/49029 и WO 01/34815.
В одном из примеров вводят ДНК, которая направляет синтез по меньшей мере частично двухцепочечного продукта(-ов) РНК, гомологичного гену мишени, который подлежит инактивации, например, такого как SDP1, TGD, пластидная GPAT, пластидная LPAAT, пластидная PAP, ген АДФ-глюкозопирофосфорилазы (АГФазы). Таким образом, ДНК содержит смысловые и антисмысловые последовательности, которые при транскрибировании в РНК могут гибридизоваться с образованием двухцепочечного участка РНК. В одном варианте осуществления, смысловая и антисмысловая последовательности отделены спейсерным участком, который содержит интрон, отщепляющийся при транскрибировании в РНК. Показано, что такое размещение приводит к более высокой эффективности сайленсинга гена (Smith et al., 2000). Двухцепочечный участок может содержать одну или две молекулы РНК, транскрибированные с одного или двух участков ДНК. Считается, что присутствие двухцепочечной молекулы запускает реакцию эндогенной системы, которая разрушает двухцепочечную РНК, а также гомологичную копию РНК, образованную геном-мишенью, эффективно снижая или устраняя активность гена-мишени.
Длина смысловой и антисмысловой последовательностей, которые гибридизуются, должна составлять для каждой по меньшей мере 19 нуклеотидов подряд, предпочтительно, по меньшей мере 50 нуклеотидов подряд, более предпочтительно, по меньшей мере 100 или по меньшей мере 200 нуклеотидов подряд. В общем, применим последовательность размером 100-1000 нуклеотидов, соответствующая участку иРНК гена-мишени. Может использоваться полноразмерная последовательность, соответствующая полному транскрипту гена. Степень идентичности смысловой последовательности целевому транскрипту (и, таким образом, также идентичности антисмысловой последовательности и последовательности, комплементарной транскрипту-мишени) должна составлять по меньшей мере 85%, по меньшей мере 90% или 95-100%. Молекула РНК может, конечно, содержать посторонние последовательности, функция которых может состоять в стабилизации молекулы. Молекула РНК может экспрессироваться под контролем промотора РНК полимеразы II или РНК полимеразы III. Примеры последнего включают промоторы тРНК или мяРНК.
Предпочтительные молекулы малой интерферирующей РНК («миРНК») содержат нуклеотидную последовательность, которая идентична приблизительно 19-25 смежным нуклеотидам иРНК-мишени. Предпочтительно, последовательность миРНК начинается с динуклеотида AA, содержит GC в количестве приблизительно 30-70% (предпочтительно, 30-60%, более предпочтительно, 40-60% и более предпочтительно, приблизительно 45-55%), и не обладает высокой степенью идентичности ни к одной нуклеотидной последовательности в геноме организма, кроме мишени, в которую она должна быть введена, например, как определяется стандартным поиском BLAST.
МикроРНК
МикроРНК (сокращенно миРНК) в общем представляют собой некодирующие молекулы РНК длиной 19-25 нуклеотидов (в растениях обычно приблизительно 20-24 нуклеотида), происходящие от крупных предшественников, которые образуют несовершенные структуры «петля-на-стебле».
миРНК связывают с дополнительными последовательностями на целевых транскриптах информационной РНК (иРНК), что обычно приводит к угнетению трансляции или разложению мишени и сайленсингу гена. Как хорошо известно в данной области, на основе природных миРНК могут быть сконструированы искусственные миРНК (имиРНК) для целей уменьшения экспрессии любого целевого гена.
Считается, что в клетках растений процессинг молекул-предшественников миРНК в значительной степени происходит в ядре. Процессинг пре-миРНК (содержащих один или более локальных двухцепочечных или «шпилечных» участков, а также обыкновенный 5' «кэп» и полиаденилированный хвост иРНК) происходит до более короткой молекулы-предшественника миРНК, которая также содержит структуру «петля-на-стебле» или конъюгацию плеч хромосомы и названа «пре-миРНК». В растениях, пре-миРНК расщепляются различными DICER-подобными ферментами (DCL), образуя дуплексы миРНК:миРНК*. Перед транспортом за пределы ядра, такие дуплексы метилируются.
В цитоплазме, цепь миРНК от спаренной миРНК:миРНК селективно встраивается в активный РНК-индуцированный комплекс сайленсинга (РИКС) для распознавания мишени. Комплексы РИКС содержат конкретное подмножество белков Argonaute, которые осуществляют специфичную в отношении последовательности репрессию гена (см., например, Millar and Waterhouse, 2005; Pasquinelli et al., 2005; Almeida and Allshire, 2005).
Косупрессия
Гены могут репрессировать экспрессию родственных эндогенных генов и/или трансгенов, уже присутствующих в геноме, где данный признак назван зависимым от гомологии сайленсингом гена. В большинстве случаев зависимый от гомологии сайленсинг гена относится к одному из двух классов - функционирующий на уровне транскрипции трансгена или действующий посттранскрипционально.
Посттранскрипциональный зависимый от гомологии сайленсинг (т. е., косупрессия) гена описывает прекращение экспрессии трансгена и родственных эндогенных или вирусных генов в трансгенных растениях. Косупрессия часто, но не всегда, возникает, если копий трансгена много, и, в общем, считается запускаемой на уровне процессинга, локализации и/или разложения иРНК. Существуют различные модели для объяснения того, как осуществляется косупрессия (см. Taylor, 1997).
Косупрессия включает введение добавочной копии гена или его фрагмента в растение, в смысловой ориентации относительно промотора, для его экспрессии. Размер смыслового фрагмента, его соответствие участкам гена-мишени, и степень идентичности его последовательности гену-мишени может определить специалист в данной области. В некоторых случаях дополнительная копия последовательности гена препятствует экспрессии растительного гена-мишени. В WO 97/20936 и EP 0465572 описаны способы осуществления способов косупрессии.
Антисмысловые полинуклеотиды
Термин «антисмысловой полинуклеотид» следует интерпретировать как обозначающий молекулу ДНК или РНК, которая комплементарна по меньшей мере части конкретной молекулы иРНК, кодирующей эндогенный полипептид, и способна препятствовать посттранскрипциональному событию, например, трансляции иРНК. Применение антисмысловых способов хорошо известно в данной области (см., например, G. Hartmann and S. Endres, Manual of Antisense Methodology, Kluwer (1999)). Применение антисмысловых способов в растениях рассмотрено Bourque (1995) и Senior (1998). Bourque (1995) перечисляет большое количество примеров того, как антисмысловые последовательности применяются в растительных системах в качестве способа инактивации гена. Bourque также заявляет, что достижение 100% ингибирования любой ферментной активности может не быть необходимым, поскольку частичное ингибирование будет более чем вероятно приводить к измеримому изменению в системе. Senior (1998) указывает, что антисмысловые способы в настоящее время представляют собой хорошо изученную технику для манипуляции экспрессией гена.
В одном варианте осуществления антисмысловой полинуклеотид гибридизуется в физиологических условиях, т. е., антисмысловой полинуклеотид (который полностью или частично является одноцепочечным) по меньшей мере способен к образованию двухцепочечного полинуклеотида с иРНК, кодирующей в клетке эндогенный полипептид, например, иРНК SDP1, TGD, пластидной GPAT, пластидной LPAAT, пластидной PAP или АГФазы, в нормальных условиях.
Антисмысловые молекулы могут включать последовательности, которые соответствуют структурным генам или для последовательностей, которые осуществляют контроль над экспрессией гена или событием укорачивания. Например, антисмысловая последовательность может соответствовать целевому кодирующему участку эндогенного гена или 5'-нетранслируемому участку (НТУ) или 3'-НТУ или их комбинации. Она может быть частично комплементарной интронным последовательностям, которые могут быть укорочены в ходе или после транскрипции, предпочтительно, только до экзонных последовательностей гена-мишени. Принимая во внимание существенную в общем дивергенцию НТУ, нацеливание данных участков обеспечивает высокую степень специфичности ингибирования гена.
Длина антисмысловой последовательности должна составить по меньшей мере 19 нуклеотидов подряд, предпочтительно, по меньшей мере 50 нуклеотидов и более предпочтительно, по меньшей мере 100, 200, 500 или 1000 нуклеотидов. Может использоваться полноразмерная последовательность, комплементарная к полноразмерному транскрипту гена. Наиболее предпочтительно, длина составляет 100-2000 нуклеотидов. Степень идентичности антисмысловой последовательности целевому транскрипту должна составлять по меньшей мере 90% и более предпочтительно, 95-100%. Антисмысловая молекула РНК, конечно, может содержать посторонние последовательности, функция которых может состоять в стабилизации молекулы.
Рекомбинантные векторы
Один вариант осуществления настоящего изобретения включает рекомбинантный вектор, который содержит по меньшей мере один полинуклеотид, раскрытый в настоящем описании, и способен доставлять полинуклеотид в клетку-хозяина. Рекомбинантные векторы включают векторы экспрессии. Рекомбинантные векторы содержат гетерологичные полинуклеотидные последовательности, т. е., полинуклеотидные последовательности, которые в природе не обнаружены смежно с полинуклеотидом, раскрытым в настоящем описании, и, предпочтительно, получены от другого вида. Вектор может представлять собой РНК или ДНК, и обычно представляет собой вирусный вектор, полученный из вируса, или плазмиду. Плазмидные векторы обычно содержат дополнительные последовательности нуклеиновой кислоты, которые включены для простоты отбора, амплификации и трансформации кассеты экспрессии в прокариотических клетках, например, pUC-полученных векторах, pGEM-полученных векторах или бинарных векторах, содержащих один или более участков T-ДНК. Дополнительные последовательности нуклеиновой кислоты включают источники репликации для автономной репликации вектора, гены селективного маркера, предпочтительно, кодирующие устойчивость к антибиотику или гербициду, уникальные множественные сайты клонирования, предусматривающие множественные сайты для вставки последовательностей нуклеиновой кислоты или кодируемых генов в конструкцию нуклеиновой кислоты, и последовательностей, которые увеличивают степень трансформации прокариотических и эукариотических (особенно растительных) клеток.
«Функционально связанный» в настоящем описании обозначает функциональное взаимоотношение двух или более фрагментов нуклеиновой кислоты (например, ДНК). Обычно, это выражение обозначает функциональное взаимоотношение элемента регуляции транскрипции (промотора) с транскрибированной последовательностью. Например, промотор функционально связан с кодирующей последовательностью полинуклеотида, описанного в настоящем описании, если он стимулирует или модулирует транскрипцию кодирующей последовательности в пригодной клетке. В общем, промоторные элементы регуляции транскрипции, которые функционально связаны с транскрибированной последовательностью, являются физически смежными с транскрибированной последовательностью, т. е., они цис-действующие. Однако, некоторые элементы регуляции транскрипции, например, энхансеры, не должны быть физически смежными или расположенными поблизости кодирующих последовательностей, транскрипцию которых они усиливают.
Если присутствует несколько промоторов, каждый промотор независимо может быть таким же или другим.
Кроме того, рекомбинантные векторы могут содержать одну или более последовательностей сигнального пептида, чтобы позволить удержание экспрессированного полипептида, описанного в настоящем описании, в эндоплазматическом ретикулуме (ER) клетки или перенос в пластиду, и/или химерные последовательности, которые приводят к экспрессии молекул нуклеиновой кислоты в виде слитых белков. Примеры пригодных сигнальных фрагментов включают любой сигнальный фрагмент, способный направлять секрецию или локализацию полипептида, описанного в настоящем описании.
Чтобы упростить идентификацию трансформантов, рекомбинантный вектор желательно должен включать ген селективного или пригодного для скрининга маркера. «Маркерный ген» обозначает ген, который передает отличный фенотип клеткам, экспрессирующим маркерный ген, и, таким образом, позволяет отличать такие трансформированные клетки от клеток, которые не содержат маркера. Ген селективного маркера обеспечивает признак, по которому может быть осуществлена «селекция» на основе устойчивости к агенту отбора (например, гербициду, антибиотику). Ген пригодного для скрининга маркера (или репортерный ген) обеспечивает признак, который можно идентифицировать посредством наблюдения или тестирования, т. е., «скрининга» (например., β-глюкуронидаза, люцифераза, GFP или другая ферментная активность, отсутствующая в нетрансформированных клетках). Примеры селективных маркеров для отбора трансформантов растения включают, но без ограничения, ген hyg, который кодирует устойчивость к гигромицину B; ген неомицинфосфотрансферазы (nptII), обеспечивающий устойчивость к канамицину, паромомицину; ген глутатион-S-трансферазы из печени крыс, обеспечивающий устойчивость к полученным из глутатиона гербицидам, например, описанный в EP 256223; гена глутаминсинтетазы, при повышенной экспрессии обеспечивающий устойчивость к ингибиторам глутаминсинтетазы, таким как фосфинотрицин, например, как описано в WO 87/05327; ген ацетилтрансферазы Streptomyces viridochromogenes, обеспечивающий устойчивость к агенту отбора фосфинотрицину, например, как описано в EP 275957; ген, кодирующий 5-енолшикимат-3-фосфат синтетазу (EPSPS), обеспечивающую переносимость N-фосфонометилглицина, например, как описано Hinchee et al. (1988); ген bar, обеспечивающий устойчивость к биалафосу, например, раскрытый в WO 91/02071; ген нитрилазы, например, bxn Klebsiella ozaenae, который обеспечивает устойчивость к бромоксинилу (Stalker et al., 1 988); ген дигидрофолатредуктазы (DHFR), обеспечивающий устойчивость к метотрексату (Thillet et al., 1988); мутантный ген ацетолактсинтетазы (АЛС), который обеспечивает устойчивость к имидазолинону, сульфонилмочевине или другим ингибирующим АЛС химическим веществам (EP 154204); модифицированный ген антранилатсинтетазы, который обеспечивает устойчивость к 5-метилтриптофану; или ген далапон дегалогеназы, который обеспечивает устойчивость к гербициду.
Предпочтительно, рекомбинантный вектор стабильно инкорпорирован в геном клетки, например, клетки растений. Соответственно, рекомбинантный вектор может содержать пригодные элементы, которые позволяют вектору инкорпорироваться в геном или хромосому клетки.
Вектор экспрессии
В настоящем описании «вектор экспрессии» представляет собой ДНК вектор, который способен к трансформации клетки-хозяина и эффективной экспрессии одного или нескольких указанных полинуклеотидов. Векторы экспрессии по настоящему изобретению содержат регуляторные последовательности, например, последовательности контроля транскрипции, последовательности контроля трансляции, источники репликации и другие регуляторные последовательности, которые совместимы с клеткой-хозяином и которые управляют экспрессией полинуклеотидаов по настоящему изобретению. В частности, векторы экспрессии по настоящему изобретению содержат последовательности контроля транскрипции. Последовательности контроля транскрипции представляют собой последовательности, которые управляют инициацией транскрипции, удлинением транскрипта и окончанием транскрипции. Особенно значимыми последовательностями контроля транскрипции являются такие, которые управляют инициацией транскрипции, например, промоторные, энхансерные, операторные и репрессорные последовательности. Выбор используемых регуляторных последовательностей зависит от организма-мишени, например, растения, и/или органа-мишени или целевой ткани. Такие регуляторные последовательности могут быть получены от любого эукариотического организма, такого как растения или вирусы растения, или могут быть химически синтезированы. Целый ряд векторов, пригодных для стабильной трансфекции растительных клеток или для получения трансгенных растений, раскрыт, например, в Pouwels et al., Cloning Vectors: A Laboratory Manual, 1985, supp. 1987, Weissbach and Weissbach, Methods for Plant Molecular Biology, Academic Press, 1989, и Gelvine et al., Plant Molecular Biology Manual, Kluwer Academic Publishers, 1990. Обычно, векторы экспрессии растений содержат, например, один или более клонированных генов растения под транскрипциональным контролем 5' и 3' регуляторных последовательностей и доминирующего селективного маркера. Кроме того, такие векторы экспрессии растений могут содержать регуляторный промоторная область (например, регуляторный участок, управляющий индуцибельной или конститутивной, регулируемой окружающей средой или стадией развития, клеточно- или тканеспецифической экспрессией), старт-сайт инициации транскрипции, сайт связывания с рибосомой, сайт терминации транскрипции и/или сигнал полиаденилирования.
Раскрыт целый ряд конститутивных промоторов, которые активны в клетках растений. Пригодные промоторы для конститутивной экспрессии в растениях включают, но без ограничения, промотор 35S вируса мозаики цветной капусты (CaMV), 35S вируса мозаики норичника (ВМН), индуцируемый светом промотор маленькой субъединицы (МСЕ) рибулозо-1,5-бисфосфат карбоксилазы, рисовый промотор цитозольной триозофосфат изомеразы, промотор аденин фосфорибозилтрансферазы Arabidopsis, промотор гена рисового актина 1, промоторы маннопин синтетазы и октопин синтетазы, промотор Adh, промотор синтетазы сахарозы, промотор комплекса R гена и промотор гена белка, связывающегося с α/βхлорофиллом. Эти промоторы использовались для создания векторов ДНК, которые экспрессируются в растениях, см., например, WO 84/02913. Все указанные промоторы использовались для создания различных видов экспрессируемых в растениях рекомбинантных ДНК векторов.
С целью экспрессии в исходных тканях растения, таких как лист, семя, корень или стебель, промоторы, используемые в настоящем изобретении, предпочтительно, демонстрируют относительно высокую экспрессию в указанных конкретных тканях. Для достижения данной цели можно выбирать из целого ряда промоторов для генов с ткане- или клеточноспецифической или -усиленной экспрессией. Примеры таких промоторов, о которых сообщалось в литературе, включают, промотор глутаминсинтетазы хлоропластов GS2 гороха, промотор фруктозо-1,6-бифосфатазы хлоропласта пшеницы, промотор ядерного фотосинтетического ST-LS1 картофеля, промотор серин/треонинкиназы и промотор глюкоамилазы (CHS) Arabidopsis thaliana. Также сообщалось, что активным в фотосинтетически активных тканях является промотор рибулозо-1,5-бисфосфат карбоксилазы восточной лиственницы (Larix laricinа), промотор гена Cab сосны, Cab6, промотор гена Cab-1 пшеницы, промотор гена Cab-1 шпината, промотор гена Cab-1R риса, промотор пируват, ортофосфат дикиназы (ПФДК) Zea mays, промотор гена Lhcb1*2 табака, промотор симпортера Suc2 Arabidopsis thaliana сахарозы-H30, и промотор для генов тилакоидного белка мембраны шпината (PsaD, PsaF, PsaE, PC, FNR, AtpC, AtpD, Cab, RbcS). Другие промоторы белков, связывающихся с α/βхлорофиллом также можно использовать в настоящем изобретении, например, промоторы гена LhcB и гена PsbP белой горчицы (Sinapis albа).
Различные промоторы растительных генов, которые регулируются в ответ на экологические, гормональные, химические и/или связанные с развитием сигналы, также можно использовать для экспрессии генов РНК-связывающего белка в клетках растения, в том числе, промоторы, регулируемые (1) нагреванием, (2) светом (например, промотор RbcS-3A гороха, промотор RbcS маиса), (3) гормонами, например, абсцизиновая кислота, (4) образованием ран (например, WunI) или (5) химическими веществами, такими как метилжасмонат, салициловая кислота, стероидные гормоны, спирт, сафенеры (WO 97/06269), или дополнительно может быть предпочтительным использование (6) специфических в отношении органа промоторов.
В настоящем описании термин «промотор, специфический в отношении запасающего органа растения» обозначает промотор, который предпочтительно, по сравнению с другими тканями растения, направляет транскрипцию гена в запасающем органе растения. С целью экспрессии в акцептирующих тканях растения, например, клубня картофельного растения, плода помидора или семени сои, рапса, хлопка, Zea mays, пшеницы, риса и ячменя, промоторам, используемым в настоящем изобретении, предпочтительно, свойственна относительно высокая экспрессия в данных конкретных тканях. Может применяться промотор β-конглицинина или другие специфичные для семени промоторы, например, промоторы напина, зеина, линина и фазеолина. Кроме того, можно использовать специфичные в отношении корней промоторы. Примером такого промотора является промотор гена кислой хитиназы. Дополнительно, экспрессия в корневой ткани могла бы быть достигнута с использованием специфических в отношении корней субдоменов промотора CaMV 35S, которые идентифицированы.
В особенно предпочтительном варианте осуществления промотор управляет экспрессией в тканях и органах, в которых имеет место биосинтез липида. Такие промоторы могут действовать на стадии развития семени, в подходящее время для модификации состава липида в семенах. Предпочтительные промоторы для специфической экспрессии в семени включают: 1) промоторы генов, кодирующих ферменты, принимающие участие в биосинтезе липида и аккумуляции в семенах, например, десатуразы и элонгазы, 2) промоторы генов, кодирующих запасаемые белки семени, и 3) промоторы генов, кодирующих ферменты, принимающие участие в биосинтезе углеводов и аккумуляции в семенах. Пригодные промоторы, специфичные в отношении семени, представляют собой: промотор гена напина масличного рапса (US 5608152), промотор Фарм. USP Vicia faba (Baumlein et al., 1991), промотор олеозина Arabidopsis (WO 98/45461), промотор фазеолина Phaseolus vulgaris (US 5504200), промотор Bce4 Brassica (WO 91/13980) или промотор легумина B4 (Baumlein et al., 1992), а также промоторы, которые приводят к специфической экспрессии в семени однодольных, таких как маис, ячмень, пшеница, рожь, рис и т. п. Следует отметить пригодные промоторы гена lpt2 или lpt1 ячменя (WO 95/15389 и WO 95/23230) или промоторы, описанные в WO 99/16890 (промоторы гена гордеина ячменя, гена глютелина риса, гена оризина риса, гена проламина риса, гена глиадина пшеницы, гена глютелина пшеницы, гена зеина маиса, гена глютелина овса, гена казирина сорго, гена секалина ржи). Другие промоторы включают описанные Broun et al. (1998), Potenza et al. (2004), US 20070192902 и US 20030159173. В одном варианте осуществления, специфический для семени промотор предпочтительно, экспрессируется в определенных частях семени, например, семядоле(-ях) или эндосперме. Примеры специфических для семядоли промоторов включают, но без ограничения, промотор FP1 (Ellerstrometal.,1996), промотор легумина гороха (Perrin et al., 2000) и промотор фитогемагглютинина боба (Perrin et al., 2000). Примеры специфических для эндоспермы промоторов включают, но без ограничения, промотор зеина-1 маиса (Chikwamba et al., 2003), промотор глютелина-1 риса (Yang et al., 2003), промотор D-гордеина ячменя (Horvath et al., 2000) и промоторы высокомолекулярного глютенина пшеницы (Alvarez et al., 2000). В дальнейшем варианте осуществления специфический для семени промотор не экспрессируется или экспрессируется только на низком уровне в зародыше и/или после того, как семя прорастает.
В другом варианте осуществления промотор, специфический для запасного органа растения, представляет собой специфический для фрукта промотор. Примеры включают, но без ограничения, промотор полигалактуроназы помидора, E8 и Pds, а также промотор аминоциклопропанкарбоксилат оксидазы (ACC) яблока (обзор см. в Potenza et al., 2004). В предпочтительном варианте осуществления промотор предпочтительно, управляет экспрессией в съедобных частях плода, например, сердцевине плода, относительно кожуры плода или семян в пределах плода.
В одном варианте осуществления, индуцибельный промотор представляет собой систему alc Aspergillus nidulans. Примеры индуцибельных систем экспрессии, которые можно использовать вместо системы Aspergillus nidulans alc, описаны в обзоре Padidam (2003) и Corrado и Karali (2009). В другом варианте осуществления индуцибельный промотор представляет собой индуцируемый сафенером промотор, например, такой как промотор ln2-1 или ln2-2 маиса (Hershey и Stoner, 1991), индуцибельный промотор сафенера представляет собой промотор GST-27 маиса (Jepson et al., 1994) или соевый промотор GH2/4 (Ulmasov et al., 1995).
В другом варианте осуществления индуцибельный промотор представляет собой промотор, индуцируемый старением, например, такой как индуцируемый старением промотор SAG (связанный со старением ген) 12 и SAG 13 Arabidopsis (Gan, 1995; Gan and Amasino, 1995) и LSC54 Brassica napus (Buchanan-Wollaston, 1994). Такие промоторы демонстрируют повышенную экспрессию в период приблизительно начала старения растительных тканей, особенно листьев.
Для экспрессии в вегетативных тканях можно использовать специфичные для листьев промоторы, такие как промоторы рибулозобифосфат карбоксилазы (RBCS). Например, гены RBCS1, RBCS2 и RBCS3A помидора экспрессируются в листьях и растущих под действием света саженцах (Meier et al., 1997). Могут использоваться промоторы рибулозобифосфат карбоксилазы, экспрессирующиеся почти исключительно в клетках мезофилла листовых пластинок и влагалищ листьев с высокими уровнями, как описано Matsuoka et al. (1994). Другой специфический для листьев промотор представляет собой легко собирающий урожай промотор гена улавливающего свет белка a/b, связывающегося с хлорофиллом (см. Shiina et al., 1997). Родственный myb промотор (Atmyb5) гена Arabidopsis thaliana, описанный Li et al. (1996), является специфическим для листьев. Промотор Atmyb5 экспрессируется в процессе развития листовых трихом, прилистников и эпидермальных клеток на краях молодой розетки и стеблевых листьях, а также в незрелых семенах. Кроме того, может использоваться листовой промотор, идентифицированный в маисе Busk et al. (1997).
В некоторых случаях, например, если LEC2 или BBM рекомбинантно экспрессируется, может быть желательным, чтобы трансген не экспрессировался с высокими уровнями. Примером промотора, который может использоваться в таких случаях, является укороченный промотор напина A, который сохраняет специфический для семени характер экспрессии, но со сниженным уровнем экспрессии (Tan et al., 2011).
5'-нетранслируемые лидерные последовательности могут происходить из промотора, выбранного для экспрессии последовательности гетерологичного гена полинуклеотида по настоящему изобретению, или могут быть гетерологичными относительно участка кодирования фермента, который должен вырабатываться, и могут быть специфически модифицированы, при желании, с целью увеличения трансляции иРНК. Обзор оптимизации экспрессии трансгенов см. в Koziel et al. (1996). 5' Нетранслируемые участки также могут быть получены из РНК вирусов растений (среди прочего, вирус мозаики табака, вирус гравировки табака, вирус карликовой мозаики маиса, вирус мозаики люцерны), из пригодных эукариотических генов, растительных генов (лидер гена белка a/b, связывающегося с хлорофиллом, пшеницы и маиса) или из синтетической последовательности гена. Настоящее изобретение не ограничивается конструкциями, в которых нетранслируемый участок получен из 5' нетранслируемой последовательности, которая сопровождает последовательность промотора. Дополнительно, лидерная последовательность может происходить от постороннего промотора или кодирующей последовательности. Лидерные последовательности, пригодные по форме и содержанию для настоящего изобретения, включают лидер Hsp70 маиса (US 5362865 и US 5859347) и элемент омега вируса мозаики табака (ВМТ).
Терминация транскрипции достигается с помощью 3' нетранслируемой последовательности ДНК, функционально связанной в векторе экспрессии с целевым полинуклеотидом. 3' Нетранслируемый участок рекомбинантной молекулы ДНК содержит сигнал полиаденилирования, функция которого в растениях состоит в присоединении аденилатных нуклеотидов к 3' концу РНК. 3' Нетранслируемый участок может быть получен из различных генов, которые экспрессируются в клетках растения. В данном случае обычно используются 3' нетранслируемые участки синтетазы нопалина, 3' нетранслируемый участок маленькой субъединицы гена РУБИСКО гороха, 3' нетранслируемый участок соевого гена запасаемого белка 7S семени. Кроме того, пригодными являются 3' транскрибированные, нетранслируемые участки, содержащие полиаденилатный сигнал индуцирующих опухоль (ИО) генов плазмиды Agrobacterium.
Рекомбинантные технологии ДНК можно использовать для улучшения экспрессии трансформированного полинуклеотида посредством манипуляции, например, эффективностью, с которой транслируются образующиеся в результате транскрипты, посредством оптимизации кодонов согласно виду клетки-хозяина или делеции последовательностей, которые дестабилизируют транскрипты, и эффективностью посттрансляционных модификаций.
Транспортные нуклеиновые кислоты
Транспортные нуклеиновые кислоты можно использовать для доставки экзогенного полинуклеотида в клетку и содержат одну, предпочтительно, две граничные последовательности и один или более целевые полинуклеотиды. Транспортная нуклеиновая кислота может кодировать или не кодировать селективный маркер. Предпочтительно, транспортная нуклеиновая кислота формирует часть бинарного вектора в клетке бактерии, где бинарный вектор дополнительно содержит элементы, которые позволяют репликацию вектора у бактерии, селекцию или поддержание бактериальных клеток, содержащих бинарный вектор. При переносе в эукариотическую клетку, компонент транспортной нуклеиновой кислоты бинарного вектора способен к интеграции в геном эукариотической клетки или пригоден для экспериментов с временной экспрессией, только с экспрессией в клетке.
В настоящем описании термин «внехромосомная транспортная нуклеиновая кислота» обозначает молекулу нуклеиновой кислоты, которая способна к переносу из бактерии, например, вида Agrobacterium, в клетку растения, например, клетку листа растения. Внехромосомная транспортная нуклеиновая кислота представляет собой генетический элемент, который известен, как элемент, способный к перемещению, с последующей интеграцией с нуклеотидной последовательностью, содержащейся в пределах его границ, в геном клетки-реципиента. В данном отношении, транспортная нуклеиновая кислота фланкирована обычно двумя «граничными» последовательностями, хотя в некоторых случаях может использоваться одна граница на одном конце, и второй конец транспортной нуклеиновой кислоты генерируется случайным образом в процессе переноса. Целевой полинуклеотид обычно размещается между левой, подобной границе, последовательностью и правой, подобной границе, последовательностью транспортной нуклеиновой кислоты. Полинуклеотид, содержащийся в пределах транспортной нуклеиновой кислоты, может быть функционально связан со множеством других промоторных и терминаторных регуляторных элементов, которые облегчают его экспрессию, т. е., транскрипцию и/или трансляцию полинуклеотида. Транспортные ДНК (Т-ДНК) видов Agrobacterium, например, Agrobacterium tumefaciens или Agrobacterium rhizogenes, и созданные человеком их варианты/мутанты, вероятно, представляют собой лучше всего описанные примеры транспортных нуклеиновых кислот. Другой пример представляет собой Р-ДНК («растительная ДНК»), которая содержит подобные границе последовательности T-ДНК из растений.
В настоящем описании «T-ДНК» обозначает T-ДНК ОИ плазмиды Agrobacterium tumefaciens или из плазмиды Ri Agrobacterium rhizogenes, или их варианты, функция которых состоит в переносе ДНК в клетки растений. T-ДНК может содержать полноразмерную T-ДНК, включая как правую, так и левую граничные последовательности, но необходимыми являются только минимальные последовательности, необходимые в цис для переноса, т. е., правая T-ДНК граничная последовательность. Т-ДНК по изобретению имеют вставленный в них где-либо между правой и левой граничными последовательностями (если они присутствуют) целевой полинуклеотид. Последовательности, кодирующие факторы, необходимые в транс для переноса T-ДНК в растительную клетку, например, гены vir, могут быть вставлены в T-ДНК или могут присутствовать на том же репликоне, что и T-ДНК, или предпочтительно, находятся в транс на совместимом репликоне в хозяине Agrobacterium. Такие «системы бинарного вектора» хорошо известны из уровня техники. В настоящем описании «П-ДНК» обозначает транспортную нуклеиновую кислоту, выделенную из генома растения или ее варианты/мутанты, созданные человеком, и содержит на каждом конце или только на одном конце подобную граничной последовательность T-ДНК.
В настоящем описании «граничная» последовательность транспортной нуклеиновой кислоты может быть выделена из выбранного организма, например, растения или бактерии, или представлять собой ее вариант/мутант, созданный человеком. Граничная последовательность способствует и облегчает перенос полинуклеотида, с которым она связана, и может облегчить его интеграцию в геном клетки-реципиента. В одном варианте осуществления, длина граничной последовательности составляет 10-80 пар оснований. Граничные последовательности T-ДНК видов Agrobacterium хорошо известны из уровня техники и включают описанные Lacroix et al. (2008).
Хотя традиционно использовались только виды Agrobacterium для переноса генов в клетки растений, в настоящее время идентифицировано/разработано большое число систем, которые функционируют подобным видам Agrobacterium способом. В последнее время несколько видов, не относящихся к Agrobacterium, были генетически модифицированы таким образом, чтобы сделать их компетентными для переноса гена (Chung et al., 2006; Broothaerts et al., 2005). Они включают вид Rhizobium NGR234, Sinorhizobium meliloti и Mezorhizobium loti.
В настоящем описании термины «трансфекция», «трансформация» и их варианты, в общем, используют взаимозаменяемо. «Трансфицированными» или «трансформированными» клетками можно манипулировать с целью введения целевого полинуклеотида(полинуклеотидов) или получения из них клеток-потомков.
Растения
Изобретение относится также к растению или его части, содержащим два или более экзогенных полинуклеотида и/или генетических модификаций как описано в настоящем описании. Термин «растение» при использовании в форме имени существительного обозначает цельные растения, тогда как термин «его часть» обозначает органы растения (например, листья, стебли, корни, цветки, плод), одиночные клетки (например, пыльцу), семя, части семени, например, зародыш, эндосперм, щиток зародыша или оболочку семени, ткань растения, например, сосудистую ткань, клетки растений и их потомки. В настоящем описании части растения включают клетки растений.
В настоящем описании термин «в растении» или «в этом растении», в контексте модификации растения означают, что модификация произошла по меньшей мере в одной части растения, включая ситуации, в которых модификация произошла во всем растению, и не исключая ситуаций, в которых модификация происходит только в одной или нескольких, но не во всех частях растения. Например, говорят, что тканеспецифический промотор экспрессируется «в растении», даже если он может экспрессироваться только в некоторых частях растения. Аналогично, «полипептид фактора транскрипции, который увеличивает экспрессию одного или нескольких гликолитических генов и/или генов биосинтеза жирных кислот в растении» означает, что повышенная экспрессия присутствует по меньшей мере в части растения.
В настоящем описании термин «растение» применим в самом широком смысле, включая любой организм из царства растений. Кроме того, он включает красные и коричневые водоросли, а также зеленые водоросли. Он включает, но без ограничения, любые виды цветковых растений, травы, сельскохозяйственной культуры или злака (например, масличная культура, маис, соя), кормового или силосного, фруктового или овощного растения, травянистого растения, деревянистого растения или дерева. Не подразумевается ограничения растения какой-либо конкретной структурой. Кроме того, он обозначает одноклеточное растение (например, микроводоросль). Термин «его часть» со ссылкой на растение обозначает растительную клетку и ее потомка, множество клеток растения, структуру, которая присутствует на любой стадии развития растения или ткани растения. Такие структуры включают, но без ограничения, листья, стебли, цветки, плоды, орехи, корни, семя, оболочку семени, зародыши. Термин «ткань растения» включает дифференцированные и недифференцированные ткани растений, в том числе, присутствующие в листьях, стеблях, цветках, плодах, орехах, корнях, семени, например, зародышевая ткань, эндосперм, ткань кожуры (например, эпидермис, перидерма), сосудистая ткань (например, ксилема, флоэма) или донная ткань (содержащая клетки паренхимы, колленхимы и/или склеренхимы), а также клетки в культуре (например, одинарные клетки, протопласты, каллюс, зародыши, и т. п.). Ткань растения может находиться in planta, в культуре органа, культуре ткани или культуре клеток.
В настоящем описании термин «вегетативная ткань» или «вегетативная часть растения» обозначает любую ткань растения, орган или часть, кроме органов для полового воспроизводства растений. В частности, органы для полового воспроизводства растений представляют собой несущие семена органы, цветки, пыльцу, плоды и семена. Вегетативные ткани и части включают по меньшей мере листья, стебли (в том числе, стрелки и побеги, за исключением головок), клубни и корни растения, за исключением цветков, пыльцы, семени, включая оболочку семени, зародыша и эндосперма, плода, включая ткани мезокарпа, несущие семя стручки и несущие семя головки. В одном варианте осуществления вегетативная часть растения представляет собой воздушную часть растения. В другом или дальнейшем варианте осуществления вегетативная часть растения представляет собой зеленую часть, например, лист или стебель.
«Трансгенное растение» или его варианты обозначают растение, которое содержит трансген, не обнаруженный в растении дикого типа такого же вида, подвида или сорта. Трансгенные растения, описанные в контексте настоящего изобретения, включают растения и их потомков, генетически модифицированных с использованием рекомбинантных способов, чтобы вызывать продукцию по меньшей мере одного полипептида, определенного в настоящем описании, в желательном растении или его части. Трансгенные части растения имеют соответствующее значение. Растение и части растения по изобретению могут содержать генетические модификации, например, мутации в генах, и их можно рассматривать как «нетрансгенные», при условии, что они лишены трансгенов.
Термины «семя» и «зерно» используют взаимозаменяемо в настоящем описании. «Зерно» обозначает зрелое зерно, например, собранное в процессе сбора урожая зерно, или зерно, которое находится на растении, но готово для сбора урожая, а также может обозначать зерно после набухания или прорастания, согласно контексту. Зрелое зерно обычно содержит менее чем приблизительно 18% влаги. В предпочтительном варианте осуществления, содержание влаги в зерне находится на уровне, который в общем расценивается как безопасный для хранения, предпочтительно, от 5% до 15%, от 6% до 8%, от 8% до 10% или от 10% до 15%. В настоящем описании «развивающееся семя» обозначает семя до состояния зрелости, обычно обнаруженное в репродуктивных структурах растения после оплодотворения или цветения, но может также обозначать семена до состояния зрелости, которые сняты с растения. Зрелое семя обычно содержит менее чем приблизительно 12% влаги
В настоящем описании термин «запасающий орган растения» обозначает часть растения, специализирующуюся на хранении энергии, например, в форме белков, углеводов, липида. Примерами запасающих органов растения являются семя, плод, клубневидные корни и клубни. Предпочтительным запасающим органом растения по изобретению является семя.
В настоящем описании термин «фенотипически нормальный» обозначает генетически модифицированное растение или его часть, например, растение, такое как трансгенное растение или запасающий орган, такой как семя, клубень или плод по изобретению, без существенного снижения способности к росту и воспроизводству, по сравнению с немодифицированным растением или его частью. Предпочтительно, биомасса, скорость роста, скорость прорастания, размер запасающего органа, размер семени и/или количество образовавшихся жизнеспособных семян составляет не менее 90% от показателей растения, не содержащего указанных генетических модификаций или экзогенных полинуклеотидов, при выращивании в идентичных условиях. Данный термин не включает признаков растения, которые могут отличаться от растения дикого типа, но которые не влияют на полноценность растения с точки зрения коммерческих целей, например, такие как фенотип балерины листьев саженца. В одном варианте осуществления, генетически модифицированное растение или его часть, которые являются фенотипически нормальными, содержат рекомбинантный полинуклеотид, кодирующий подавляющий сайленсинг, функционально связанный с промотором, специфическим для запасающего органа растения, и обладают способностью к росту или воспроизводству, по существу такой же как соответствующее растение или его часть, не содержащие указанного полинуклеотида.
Растения проходят серию стадий роста от посева семени, прорастания и появления сеянца, до цветения, завязывания семян, физиологической зрелости и, в конечном счете, старения. Эти стадии хорошо известны и могут быть легко определены, например, для растений Sorghum, следующим образом. Принимая сутки, на которые сеянец впервые появляется над грунтом, за сутки 0, вегетативную стадию роста определяют в настоящем описании как от 10 суток до начала цветения на приблизительно 60-70 сутки, и физиологическая зрелость достигается на приблизительно 100 сутки, в зависимости от условий внешней среды. Вегетативная стадия включает стадию трубчатого листа от приблизительно 45 суток до первого цветения. Трубчатый лист представляет собой последний лист, образующийся у растения, из которого появляется метелка (верхушка). «Стадию трубчатого листа» определяют как период от полного появления трубчатого листа до начала цветения.
В настоящем описании «начало цветения» или «наступление цветения» со ссылкой на растение обозначает время раскрытия первого цветка на растении или время начала пыления.
В настоящем описании термин «завязывание семян» со ссылкой на несущее семена растение обозначает время, когда первое семя растения достигает зрелости. Обычно это можно определить по цвету семени или содержанию в нем влаги, как хорошо известно в данной области.
Как используют в настоящем описании, термин «зрелый» применительно к листу растения, означает, что он достиг полного размера, но не начал проявлять признаки старения или гибели, такие как пожелтение и/или увядание. Специалист в данной области может легко определить, можно ли лист конкретного растения рассматривать как зрелый.
В настоящем описании термин «старение» со ссылкой на цельное растение обозначает конечную стадию развития растения, которая следует за завершением роста, обычно после того, как воздушная биомасса или высота растения достигает максимума. Старение начинается, когда воздушная биомасса растения достигает максимума, и ее количество начинает уменьшаться, и в общем заканчивается с гибелью большинства тканей растения. В ходе данной стадии растение мобилизует и рециклизует клеточные компоненты из листьев и других частей, которые накапливались в процессе роста, в другие части, чтобы завершить репродуктивное развитие. Старение представляет собой сложный, регулируемый процесс, в который вовлечена экспрессия новых генов или повышенная экспрессия некоторых генов. Часто, некоторые части растения стареют, в то время как другие части того же растения продолжают расти. Таким образом, относительно листа растения или другого зеленого органа, термин «старение» в настоящем описании обозначает время, когда количество хлорофилла в листе или органе начинает снижаться. Старение обычно сопровождается обезвоживанием листа или органа, по большей части в последней стадии старения. Старение обычно можно определить по изменению цвета листа с зеленого по направлению к желтому и, в конечном счете, к коричневому окрашиванию после полного обезвоживания. Считается, что клеточное старение лежит в основе старения растения и органов.
Растения, описанные или предусмотренные для применения в практике настоящего изобретения, включают однодольные или двудольные. В предпочтительных вариантах осуществления, растения по настоящему изобретению представляют собой сельскохозяйственные растения (например, злаковые и зернобобовые, маис, пшеницу, различные виды картофеля, рис, сорго, пшено, маниок, ячмень), или другие растения из семейства бобовых, такие как соя, фасоль или горох. Растения могут выращиваться для производства съедобных корней, клубней, листьев, стеблей, цветков или плодов. Растения могут быть овощными растениями, вегетативные части которых используются в пищу. Растения по изобретению могут представлять собой: Acrocomia aculeata (макаубская пальма), Arabidopsis thaliana, Aracinis hypogaea (арахис), Astrocaryum murumuru (мурумуру), Astrocaryum vulgare (тукума), Attalea geraensis (атталия), Attalea humilis (американская масличная пальма), Attalea oleifera (андайя), Attalea phalerata (урикури), Attalea speciosa (бабассу), Avena sativa (овес), Beta vulgaris (сахарная свекла), виды Brassica, такие как Brassica carinata, Brassica juncea, Brassica napobrassica, Brassica napus (рапс), Camelina sativa (ложный лен), Cannabis sativa (конопля), Carthamus tinctorius (сафлор красильный), Caryocar brasiliense (пекви), Cocos nucifera (кокос), Crambe abyssinica (абиссинская капуста), Cucumis melo (дыня), Elaeis guineensis (африканская пальма), Glycine max (соя), Gossypium hirsutum (хлопчатник), виды Helianthus, такие как Helianthus annuus (подсолнечник), Hordeum vulgare (ячмень), Jatropha curcas (лечебный орех), Joannesia princeps (лещина арара), виды Lemna (ряска), такие как Lemna aequinoctialis, Lemna disperma, Lemna ecuadoriensis, Lemna gibba (ряска горбатая), Lemna japonica, Lemna minor, Lemna minuta, Lemna obscura, Lemna paucicostata, Lemna perpusilla, Lemna tenera, Lemna trisulca, Lemna turionifera, Lemna valdiviana, Lemna yungensis, Licania rigida (ойтика), Linum usitatissimum (лен), Lupinus angustifolius (люпин), Mauritia flexuosa (пальма бурити), Maximiliana maripa (пальма инайя), виды Miscanthus, такие как Miscanthus x giganteus и Miscanthus sinensis, виды Nicotiana (табак), такие как Nicotiana tabacum или Nicotiana benthamiana, Oenocarpus bacaba (бакаба-до-азейте), Oenocarpus bataua (патайя), Oenocarpus distichus (бакаба-де-лек), виды Oryza (рис), такие как Oryza sativa и Oryza glaberrima, Panicum virgatum (просо прутьевидное), Paraqueiba paraensis (марь), Persea amencana (авокадо), Pongamia pinnata (индийский бук), Populus trichocarpa, Ricinus communis (клещевина), виды Saccharum (сахарный тростник), Sesamum indicum (кунжут), Solanum tuberosum (картофель), виды Sorghum, такие как Sorghum bicolor, Sorghum vulgare, Theobroma grandiforum (какао), виды Trifolium, Trithrinax brasiliensis ((бразильская игольчатая пальма), виды Triticum (пшеница), такие как Triticum aestivum, Zea mays (кукуруза), люцерна (Medicago sativa), рожь (Secale cerale), сладкий картофель (Lopmoea batatus), маниок (Manihot esculenta), кофе (виды Cofea), ананас (Anana comosus), цитрусовое дерево (виды Citrus), какао (Theobroma cacao), чай (Camellia senensis), банан (виды Musa), авокадо (Persea americana), инжир (Ficus casica), гуава (Psidium guajava), манго (Mangifer indica), олива (Olea europaea), папайя (Carica papaya), кешью (Anacardium occidentale), макадамия (Macadamia intergrifolia) и миндаль (Prunus amygdalus).
В одном варианте осуществления, растение не относится к виду Nicotiana.
Другие предпочтительные растения включают травы C4, например, в дополнение к упомянутым выше, Andropogon gerardi, Bouteloua curtipendula, B. gracilis, Buchloe dactyloides, Schizachyrium scoparium, Sorghastrum nutans, Sporobolus cryptandrus; травы C3, например, Elymus canadensis, фасоль Lespedeza capitata и Petalostemum villosum, полукустарник Aster azureus; и древесные растения, например, Quercus ellipsoidalis и Q. macrocarpa. Другие предпочтительные растения включают травы C3.
В предпочтительном варианте осуществления растение представляет собой покрытосеменное растение.
В одном варианте осуществления, растение представляет собой растение масличной культуры, предпочтительно, сельскохозяйственной масличной культуры. В настоящем описании «растение масличной культуры» представляет собой вид растения, используемый для коммерческого получения липида из семян растения. Растение масличной культуры может представлять собой, например, масличный рапс (например, рапс), маис, подсолнечник, сафлор красильный, сою, сорго, лен (семя льна) или сахарную свеклу. К тому же, растение масличной культуры может быть другим представителем Brassica, хлопчатником, арахисом, маком, брюквой, горчицей, клещевиной, кунжутом, сафлором, Jatropha curcas или орехоносным растением. Растение может вырабатывать высокие уровни липида в плоде, например, олива, масличная пальма или кокосовая пальма. Садовые растения, к которым может быть применено настоящее изобретение, представляют собой салат, эндивий или растения вида Brassica, включая капусту, брокколи или цветную капусту. Настоящее изобретение может быть применено к табаку, тыквам, моркови, землянике, помидору или перцу.
В предпочтительном варианте осуществления, растение является членом семейства Fabaceae (или Leguminosae), таким как клевер, горох, люцерна посевная, фасоль, чечевица, люпин, мескито, кэроб, фасоль, и/или членом семейства Poaceae, таким как кукуруза, сорго, пшеница, ячмень и овес. В особенно предпочтительном варианте осуществления, растение представляет собой клевер, кукурузу или сорго, каждое из которых можно использовать, в частности, для фуража или грубого корма для животных.
В предпочтительном варианте осуществления трансгенное растение является гомозиготным по всем и каждому генам, которые введены (трансгенам), таким образом, что его потомок, не отщепляется от желательного фенотипа. Кроме того, трансгенное растение может являться гетерозиготным по введенному трансгену(трансгенам), предпочтительно, однородно гетерозиготным по трансгену, например, в потомстве F1, выращенном из гибридного семени. Такие растения могут обеспечивать преимущества, например, силу гибрида, хорошо известную в данной области.
Трансформация растений
Трансгенные растения могут быть получены с использованием способов, известных из уровня техники, например, раскрытых в общих чертах в Slater et al., Plant Biotechnology - The Genetic Manipulation of Plants, Oxford University Press (2003), и Christou and Klee, Handbook of Plant Biotechnology, John Wiley and Sons (2004).
В настоящем описании термин «стабильно трансформирующий», «стабильно трансформированный» и его варианты обозначают интеграцию полинуклеотида в геном клетки таким образом, что он передается клеткам-потомкам в процессе деления клетки, без необходимости в положительной отбора на предмет их присутствия. Стабильные трансформанты или их потомки могут быть идентифицированы любыми средствами, известными из уровня техники, например, Саузерн-блоттингом на хромосомной ДНК или гибридизацией геномной ДНК in situ, которые позволяют осуществить их отбор.
Опосредованный Agrobacterium перенос представляет собой широко применимую систему для введения генов в клетки растений, поскольку ДНК может быть введена в клетки цельных тканей растения, органов растения или эксплантов в культуре ткани, для временной экспрессии или стабильной интеграции ДНК в геном растительной клетки. Например, можно применять способы с погружением цветков (в полевых условиях). Применение опосредованных Agrobacterium векторов интеграции в растения с целью введения ДНК в клетки растений хорошо известно в данной области. Участок ДНК для переноса определяется последовательностями границы, и перенесенная ДНК (T-ДНК) обычно вставляется в геном растения. Данный способ является способом выбора вследствие простой и надежной природы переноса гена.
Способы ускорения, которые можно использовать, включают, например, баллистическую трансфекцию и т. п. Одним из примеров способа доставки трансформирующих молекул нуклеиновых кислот в клетки растений является баллистическая трансфекция. Данный способ рассмотрен Yang et al., Particle Bombardment Technology for Gene Transfer, Oxford Press, Oxford, England (1994). Небиологические частицы (микрочастицы), которые могут быть покрыты нуклеиновыми кислотами и доставлены в клетки, например, незрелых эмбрионов, толкающей силой. Примеры частиц включают состоящие из вольфрама, золота, платины и т.п.
В другом способе пластиды могут быть стабильно трансформированы. Способы, описанные для трансформации пластиды в высших растениях, включают доставку с помощью пистолета частицы ДНК, содержащей селективный маркер, и нацеливание ДНК на геном пластиды посредством гомологичной рекомбинации (US 5451513, US 5545818, US 5877402, US 5932479 и WO 99/05265). Дополнительно, можно использовать другие способы трансформации клетки, которые включают, но без ограничения, введение ДНК в растения прямым переносом ДНК в пыльцу, прямой инъекцией ДНК в репродуктивные органы растения или прямой инъекцией ДНК в клетки незрелых зародышей, с последующей регидратацией высушенных зародышей.
Регенерация, развитие и культивирование растений из единичных трансформантов протопласта растения или из различных трансформированных эксплантов хорошо известны из уровня техники (Weissbach et al., In: Methods for Plant Molecular Biology, Academic Press, San Diego, Calif., (1988)). Такая регенерация и процесс роста обычно включают стадии отбора трансформированных клеток, культивирования таких индивидуализированных клеток через обыкновенные стадии зародышевого развития, включая стадию укоренившегося сеянца. Трансгенные зародыши и семена регенерируют подобным образом. Полученные трансгенные укоренившиеся побеги в дальнейшем пересаживают в пригодную среду для роста растения, например, грунт.
Развитие или регенерация растений, содержащих чужеродный, экзогенный ген, хорошо известны из уровня техники. Предпочтительно, регенерирующиеся растения самоопыляются, чтобы обеспечить гомозиготные трансгенные растения. В другом случае, пыльцу, полученную от регенерирующих растений, скрещивают с выращенными из семени растениями важных с агрономической точки зрения линий. С другой стороны, пыльца растений таких важных линий используется для опыления регенерированных растений. Трансгенное растение по настоящему изобретению, содержащее желательный полинуклеотид, культивируют с использованием способов, хорошо известных специалисту в данной области.
Чтобы подтвердить присутствие трансгенов в трансгенных клетках и растениях, может быть осуществлена амплификация полимеразной цепной реакцией (ПЦР) или анализ способом Саузерн-блоттинга, с использованием способов, известных специалистам из уровня техники. Продукты экспрессии трансгенов могут быть обнаружены любым из множества путей, в зависимости от природы продукта, которые включают Нозерн-блоттинг гибридизацию, Вестерн-блоттинг и ферментный анализ. Как только трансгенные растения получены, они могут выращиваться для получения тканей или частей растений, содержащих желательный фенотип. Урожай ткани растения или частей растений может быть собран, и/или семена могут быть собраны. Семя может служить источником для выращивания дополнительных растений с тканями или частями, обладающими желательными характеристиками. Предпочтительно, урожай вегетативных частей растений собирают в то время, когда выход неполярных липидов находится на самом высоком уровне. В одном варианте осуществления урожай вегетативных частей растений собирают во время, близкое к цветению или после начала цветения. Предпочтительно, части растения собирают в период приблизительно начала старения, на который обычно указывает пожелтение и высыхание листьев.
Трансгенные растения, полученные с использованием Agrobacterium или других способов трансформации, обычно содержат единственный генетический локус на одной хромосоме. Такие трансгенные растения могут обозначаться как гемизиготные по дополнительному гену(генам). Более предпочтительным является трансгенное растение, гомозиготное по дополнительному гену(генам), т. е. трансгенное растение, которое содержит два дополнительных гена, по одному гену в одинаковых локусах на каждой хромосоме из пары хромосом. Гомозиготное трансгенное растение может быть получено посредством самооплодотворения гемизиготного трансгенного растения, проращиванием части полученных семян и анализом полученных растений на предмет присутствия представляющего интерес гена.
Кроме того, необходимо понимать, что два различных трансгенных растения, которые содержат два независимо разделенных экзогенных гена или локуса, могут быть скрещены (получен гибрид), с целью получения потомства, содержащего оба набора генов или локусов. Самоопыление подходящего потомка F1 может давать растения, гомозиготные по обоим экзогенным генам или локусам. Обратное скрещивание с материнским растением и скрещивание с нетрансгенным растением также предусмотрены, как и вегетативное размножение. Подобным образом, трансгенное растение может быть скрещено со вторым растением, содержащим генетическую модификацию, такую как мутантный ген, с идентификацией потомства, содержащего как трансген, так и генетическую модификацию. Описание других способов разведения, которые обычно применяются для различных признаков и сельскохозяйственных культур, можно найти в Fehr, In: Breeding Methods for Cultivar Development, Wilcox J. ed., American Society of Agronomy, Madison Wis. (1987).
TILLING
В одном варианте осуществления TILLING (Индуцированные Нацеливанием Локальные Повреждения В Геномах, TILLING) может применяться для получения растений, в которых эндогенные гены содержат мутацию, например, гены, кодирующие полипептид SDP1 или TGD, TST, пластидную GPAT, пластидную LPAAT, фосфатазу фосфатидиновой кислоты (PAP) или комбинацию двух или более из указанных компонентов. На первой стадии введение мутаций, например новых модификаций одинарных пар оснований, осуществляют в популяции растений посредством обработки семян (или пыльцы) химическим мутагеном, с последующим разведением растений до поколения, в котором мутации будут устойчиво наследоваться. ДНК извлекают и хранят семена всех членов популяции, чтобы создать ресурс, который может быть доступен повторно через некоторое время. Для анализа TILLING можно применить гетеродуплексные способы с использованием специфических эндонуклеаз, с целью выявления однонуклеотидного полиморфизма (SNP). В качестве альтернативы, секвенирование ДНК следующего поколения из пулов мутантных растений можно применять для идентификации мутаций в выбранном гене. Обычно, достигается частота мутаций один мутант на 1000 растений в популяции мутантных растений. С применением данного подхода может быть осуществлен скрининг многих тысяч растений для идентификации любого индивидуума с модификацией единственного основания, а также небольшими инсерциями или делециями (1-30 пар оснований) в любом гене или конкретном участке генома. TILLING дополнительно описана Slade and Knauf (2005) и Henikoff et al. (2004).
В дополнение к возможности эффективного обнаружения мутаций, высокопроизводительная технология TILLING идеальна для обнаружения природного полиморфизма. Таким образом, детальное исследование неизвестной гомологичной ДНК, образующей гетеродуплекс с известной последовательностью, выявляет количество и расположение полиморфных сайтов. Идентифицированы нуклеотидные модификации, а также инсерции и делеции небольшого размера, в том числе, по меньшей мере некоторые полиморфизмы количества повторов. Указанный признак получил название Ecotilling (Comai et al., 2004).
Редактирование генома с использованием сайт-специфических нуклеаз
Редактирование генома с использованием сконструированных нуклеаз, таких как направляемые РНК эндонуклеазы или нуклеазы ДНК, состоящие из доменов связывания ДНК, специфических для последовательности, слитых с неспецифическим модулем расщепления ДНК. Такие сконструированные нуклеазы дают возможность осуществлять эффективные и точные генетические модификации посредством индукции направленных разрывов двухцепочечной ДНК, которые побуждают эндогенные клеточные механизмы восстановления ДНК клетки к восстановлению индуцированного разрыва. Такие механизмы включают, например, подверженное ошибкам негомологичное соединение концов (NHEJ) и направляемое гомологией восстановление (HDR).
В присутствии донорской плазмиды с удлиненными гомологичными плечами, HDR может приводить к введению одного или нескольких трансгенов для коррекции или замены существующих генов. В отсутствии донорской плазмиды, NHEJ-опосредованное восстановление дает незначительные инсерционные или делеционные мутации мишени, которые вызывают разрушение гена.
Сконструированные нуклеазы, пригодные для способов по настоящему изобретению, включают цинк-пальцевые нуклеазы (ZFN), подобные активатору транскрипции (TAL) эффекторные нуклеазы (TALEN) и нуклеазы типа CRISPR/Cas9, и родственные нуклеазы.
Обычно кодирующие нуклеазу гены доставляются в клетки плазмидной ДНК, вирусными векторами или транскрибированной in vitro мРНК.
Цинк-пальцевая нуклеаза (ZFN) содержит домен связывания с ДНК и домен расщепления ДНК, где домен связывания с ДНК состоит по меньшей мере из одного «цинкового пальца» и функционально связан с доменом расщепления ДНК. Домен связывания с ДНК цинкового пальца расположен на N-конце белка, а домен расщепления ДНК расположен на C-конце указанного белка.
ZFN должна содержать по меньшей мере один «цинковый палец». В предпочтительном варианте осуществления ZFN должна содержать по меньшей мере три «цинковых пальца», чтобы обладать достаточной специфичностью и быть пригодной для направленной генетической рекомбинации в клетке- или организме-хозяине. Обычно, специфичность ZFN, содержащей более чем три «цинковых пальца», будет постепенно увеличиваться с наличием каждого дополнительного «цинкового пальца».
Домен «цинкового пальца» может происходить из любого класса или вида «цинковых пальцев». В конкретном варианте осуществления домен «цинкового пальца» включает Cis2His2 тип «цинкового пальца», который в самом общем виде представлен, например, факторами транскрипции «цинкового пальца» TFIIIA или Sp1. В предпочтительном варианте осуществления домен «цинкового пальца» включает три «цинковых пальца» типа Cis2His2. Распознавание ДНК и/или специфичность связывания ZFN могут быть модифицированы с целью достижения целевой генетической рекомбинации на каком-либо выбранном сайте клеточной ДНК. Такая модификация может быть достигнута с использованием известных способов молекулярной биологии и/или химического синтеза (см., например, Bibikova et al., 2002).
Домен расщепления ДНК ZFN получают из класса неспецифических доменов расщепления ДНК, например домен расщепления ДНК рестрикционного фермента типа II, например, FokI (Kim et al., 1996). Другие пригодные эндонуклеазы могут содержать, например, HhaI, HindIII, Nod, BbvCI, EcoRI, BglI и AlwI.
Подобная активатору транскрипции (TAL) эффекторная нуклеаза (TALEN) содержит домен связывания с ДНК эффектора TAL и домен эндонуклеазы.
Эффекторы TAL представляют собой белки патогенных бактерий растения, которые впрыскиваются патогеном в растительную клетку, где они перемещаются к ядру и функционируют, как факторы транскрипции для активизации специфических генов растения. Первичная аминокислотная последовательность эффектора TAL диктует нуклеотидную последовательность, с которой он связывается. Таким образом, для эффекторов TAL могут быть предсказаны сайты-мишени, и эффекторы TAL могут быть сконструированы и сгенерированы для целей связывания с конкретными нуклеотидными последовательностями.
Слитые с эффектором TAL кодирующие последовательности нуклеиновых кислот представляют собой последовательности, кодирующие нуклеазу или часть нуклеазы, обычно неспецифический домен расщепления рестрикционной эндонуклеазы типа II, например, FokI (Kim et al., 1996). Другие пригодные эндонуклеазы могут содержать, например, HhaI, HindIII, Nod, BbvCI, EcoRI, BglI и AlwI. Тот факт, что некоторые эндонуклеазы (например, FokI) функционируют только в виде димеров, может быть с выгодой использован для повышения специфичности эффектора TAL в отношении мишени. Например, в некоторых случаях каждый мономер FokI может быть слит с последовательностью эффектора TAL, которая распознает другую последовательность мишени ДНК, и только в случае, когда два сайта распознавания находятся в непосредственной близости, неактивные мономеры сближаются настолько, чтобы совместно образовать функционирующий фермент. С включением необходимости в связывании с ДНК для активизации нуклеазы, может быть создан высокоспецифический в отношении сайта рестрикционный фермент.
Специфичная для последовательности TALEN может распознавать конкретную последовательность в пределах предварительно выбранной нуклеотидной последовательности-мишени, присутствующей в клетке. Таким образом, в некоторых вариантах осуществления нуклеотидная последовательность-мишень может быть просканирована на предмет наличия сайтов для распознавания нуклеазой, и конкретная нуклеаза может быть выбрана на основании последовательности мишени. В других случаях, может быть сконструирована TALEN, нацеленная на конкретную клеточную последовательность.
Редактирование генома с использованием программируемых РНК-направляемых ДНК эндонуклеаз
В отличие от сайт-специфических нуклеаз, описанных выше, система кластеризованных, коротких палиндромных повторов с регулярными пропусками (CRISPR, CRISP)/Cas предлагает альтернативу ZFN и TALEN для индукции направленных генетических модификаций посредством направляемого РНК расщепления ДНК.
Системы CRISPR зависят от CRISPR РНК (ккРНК) и трансактивирующей химерной РНК (траккРНК) для осуществления специфичного для последовательности расщепления ДНК. Существует три вида систем CRISPR/Cas: в системах II типа Cas9 служит направляемой РНК ДНК эндонуклеазой, которая расщепляет ДНК при распознавании мишени ккРНК-траккРНК. Основание CRISPR РНК спаривается с траккРНК с образованием структуры из двух РНК, которая направляет Cas9 эндонуклеазы к комплементарным сайтам ДНК для расщепления.
Система CRISPR может быть доставлена в клетки растений посредством совместной доставки плазмид, экспрессирующих эндонуклеазу Cas и необходимые компоненты ккРНК. Эндонуклеаза Cas может быть превращена в никазу, чтобы обеспечить дополнительный контроль над механизмом восстановления ДНК (Cong et al., 2013).
CRISPR обычно представляют собой короткие, частично палиндромные последовательности размером 24-40 пар оснований, содержащие внутренние и концевые перевернутые повторы размером до 11 пар оснований. Хотя обнаружены изолированные элементы, в общем они организованы в кластеры (в количестве до приблизительно 20 или более на геном) повторяющихся фрагментов, разделенных уникальными последовательностями размером 20-58 пар оснований. В общем, CRISPR являются гомогенными в пределах конкретного генома, и большинство из них идентичны. Однако, существуют примеры гетерогенности, например, в Archaea (Mojica et al., 2000).
Пищевые продукты
Настоящее изобретение относится к композициям, которые можно использовать в качестве пищевых продуктов. Для целей настоящего изобретения, «пищевые продукты» включают любую пищу или препарат, который служит для употребления животным (включая человека) и который служит для питания или построения тканей, или поставки энергии, и/или для сохранения, восстановления или поддержания адекватного пищевого статуса или метаболической функции. Пищевые продукты по изобретению включают питательные композиции для грудных детей и/или детей младшего возраста.
Как используют в настоящем описании, термин «животное» относится к любому эукариотическому организму, способному проглатывать полученный из растения материал. В одном варианте осуществления, животное представляет собой жвачное животное (крупный рогатый скот, овец, коз и т.д.). Альтернативно, животное представляет собой нежвачное животное. В одном варианте осуществления, животное представляет собой млекопитающее. В одном варианте осуществления, животное представляет собой человека. В одном варианте осуществления, животное представляет собой сельскохозяйственное животное, такое как, но без ограничения, крупный рогатый скот, козы, овцы, свиньи, лошади, домашняя птица, такая как куры, и т.п. В одном варианте осуществления, крупный рогатый скот представляет собой молочный крупный рогатый скот или мясной крупный рогатый скот. В другом варианте осуществления, животное представляет собой рыбу, например, рыбу, выведенную с использованием аквакультуры, включая, но без ограничения, лосося, форель, карпа, окуня, леща, палтуса, камбалу, молочную рыбу, кефаль, групера, лиманду, морского окуня, треску, пикшу, ложного палтуса, сома, гольца, сига, белугу, линя, плотву, щуку, судака, желтохвоста, тиляпию, угря или тропическую рыбу (такую как пресноводная, солоноватоводная и солоноводная тропическая рыба). Животное может представлять собой ракообразное, такое как, но без ограничения, криль, промысловые моллюски, креветка (включая большую креветку), краб и омар.
Пищевые продукты по изобретению могут включать, например, растение или его часть, такую как вегетативная часть растения по изобретению, вместе с пригодным носителем(носителями). Термин «носитель» используют в его самом широком смысле для включения любого компонента, который может иметь или может не иметь питательную ценность. Как понятно специалисту в данной области, носитель должен является пригодным для использования (или используемым в достаточно низкой концентрации) в пищевом продукте, так что он не оказывает неблагоприятного эффекта на организм, который потребляет пищевой продукт. Пищевые продукты могут содержать части растений, собранные и впоследствии переработанные или обработанные, например, посредством рубки, нарезки, сушки, прессования или гранулирования частей растений, в форму, пригодную для употребления животным, или измененные посредством таких способов, как сушка или ферментация, для получения сена или силоса.
Пищевой продукт по настоящему изобретению содержит липид и/или белок, полученный напрямую или опосредованно с использованием способов, растений или их частей, описанных в настоящем описании. Композиция может находиться либо в твердой или жидкой форме. Кроме того, композиция может включать съедобные питательные макровещества, витамины и/или минералы, в количествах, желательных для конкретного использования. Количества этих ингредиентов можно менять в зависимости от того, предназначена ли композиция для использования нормальными индивидуумами или для использования индивидуумами, имеющими специализированные потребности, такими как индивидуумы, страдающие метаблическими нарушениями, и т.п.
Примеры пригодных носителей с питательной ценностью включают, но без ограничения, питательные макровещества, такие как съедобные жиры, углеводы и белки. Примеры съедобных жиров включают, но без ограничения, кокосовое масло, масло бурачника, грибное масло, масло из почек черной смородины, соевое масло и моно- и диглицериды. Примеры таких углеводов включают, но без ограничения, глюкозу, съедобную лактозу и гидролизованный крахмал. Кроме того, примеры белков, которые можно использовать в пищевой композиции по изобретению, включают, но без ограничения, соевые белки, подвергнутую электродиализу молочную сыворотку, подвергнутое электродиализу обезжиренное молоко, молочную сыворотку или гидролизаты этих белков.
Применительно к витаминам и минералам, следующие можно добавлять в композиции пищевого продукта по настоящему изобретению, кальций, фосфор, калий, натрий, хлорид, магний, марганец, железо, медь, цинк, селен, иод и витамины A, E, D, C и комплекса B. Можно добавлять также другие такие витамины и минералы.
Композицию пищевого продукта по настоящему изобретению можно также добавлять в пищу, даже когда дополнение диеты не требуется. Например, композицию можно добавлять в пищу любого типа, включая, но без ограничения, маргарин, масло, виды сыра, молоко, йогурт, шоколад, конфеты, закуски, салатные масла, кулинарные масла, кулинарные жиры, мясные продукты, рыбные продукты и напитки.
Кроме того, материал, полученный produced в соответствии с настоящим изобретением, можно также использовать в качестве пищевых добавок для животных для изменения состава жирных кислот в ткани или молоке животного на состав, более желательный для употребления человеком или животным, или для уменьшения продукции метана у жвачных животных. Кроме того, пищевые продукты по изобретению можно использовать в аквакультуре для увеличения уровней жирных кислот и питательной ценности в рыбе для употребления человеком или животным.
Предпочтительными пищевыми продуктами по изобретению, являются растения, семена и другие части растений, такие как such листья, плоды и стебли, которые можно использовать непосредственно в качестве пищи или корма для человека или других животных. Например, животных можно пасти непосредственно среди таких растений, растущих в поле, или их можно кормить более точно измеренными количествами при контролируемом кормлении. Изобретение относится к использованию таких растений и частей растений в качестве пищи для увеличения уровней полиненасыщенных жирных кислот у человека и других животных.
Для употребления не относящимися к человеку животными, пищевой продукт может находиться в любой пригодной для этого форме, такой как, но без ограничения, силос, сено или подножный корм, растущий в поле. В одном варианте осуществления, пищевой продукт, не предназначенный для употребления человеком, представляет собой бобовое растение или его часть, являющееся членом семейства Fabaceae (или Leguminosae), такие как клевер, горох, люцерна посевная, фасоль, чечевица, люпин, мескито, кэроб, соя и арахис.
В одном варианте осуществления, животное находится в загоне для кормления скота и/или под навесом.
В одном варианте осуществления, растение или его фракция составляет по меньшей мере приблизительно 5%, по меньшей мере приблизительно 10%, по меньшей мере приблизительно 50%, по меньшей мере приблизительно 75%, по меньшей мере приблизительно 90% или весь пищевой продукт.
Силос
Как используют в настоящем описании, «силос» представляет собой фураж с относительно высоким содержанием влажности, который получают и хранят способом, называемым силосованием, и которым, как правило, кормят крупный рогатый скот, овец или других жвачных. В ходе периода хранения, углеводы, липиды и белки в растительном материале подвергаются ферментации с образованием органических кислот, или окислительному разложению, или и тому, и другому. Как растительный материал после сбора, так и растительные материалы после ферментации включены в силос, как этот термин используют в настоящем описании. Силос, как правило, получен из кормовой посевной травы, такой как маис, сорго, овес или другие злаки, или из смешанных трав и бобовых из подножного корма, таких как люцерна или клевер, с использованием зеленых, надземных частей растений. Силос получают либо посредством помещения вегетативного материала растений (обычно, биомассы всей надземной части растения, которая может включать репродуктивные ткани) в силосную яму или силосную башню, или другие средства для хранения, и их прессование, чтобы оставить настолько мало воздуха, насколько возможно, в растительном материале. Кислород убирают до некоторой степени посредством его покрытия листами пластика или посредством плотного заворачивания растительного материала в пластиковую пленку (упаковку в тюки) для уменьшения притока воздуха. Силос получают из растительного материала с подходящим содержанием влажности, как правило, приблизительно 50%-60% от массы в свежем состоянии, в зависимости от используемых способов хранения и степени сжатия, и количества воды, которое может быть потеряно при хранении, но не превышающего 75%. В случае сорго и кукурузы, сбор начинают, когда влажность целого растения находится на подходящем уровне, в идеале, за несколько суток до их созревания. В случае сельскохозяйственных культур типа подножного корма, растения скашивают и позволяют увядание в течение суток или пока содержание влажности не снизится до подходящего уровня. В идеале, сельскохозяйственные культуры скашивают в высшей точке цветения и закладывают на хранение в силосную яму или силосную башню на сутки их нарезки. При сборе или после, растительный материал шинкуют или рубят посредством комбайна на фрагменты, как правило, длиной приблизительно 1-5 см. Растительный материал можно помещать, в больших копнах, на землю и прессовать для уменьшения количества воздуха, затем накрывать пластиком, или можно помещать в силосную башню. Альтернативно, растительный материал можно упаковывать в тюки в пластиковой обертке для удаления воздуха, что, как правило, требует более низкого содержания влажности приблизительно 30-40%, но все еще является слишком влажным для хранения в форме сухого сена.
Нарезанный или нарубленный растительный материал при хранении подвергается в основном анаэробной ферментации, которая начинается приблизительно через 48 часов после заполнения силосной ямы или силосной башни. Процесс ферментации переводит сахара и другие углеводы, такие как гемицеллюлоза, в органические кислоты, в основном, уксусную, пропионовую, молочную и масляную кислоты. Ферментация начинается после потребления захваченного кислорода и в основном заканчивается через приблизительно две недели хранения, или может продолжаться приблизительно более длительных периодов. Когда растительный материал плотно упакован, поступление кислорода ограничено, и ферментация приводит к разложению углеводов, некоторых липидов и белков в материале до органических кислот. Этот продукт назван кислым силосом. Если, с другой стороны, фураж упакован более свободно, основной реакцией является окисление, которое происходит более быстро, и температура повышается. Если массу прессуют, когда температура составляет 60-75C, реакция останавливается, и в результате образуется сладкий силос. Ферментации можно способствовать посредством инокуляции специфических микроорганизмов, таких как молочнокислые бактерии, для ускорения ферментации или улучшения полученного силоса, например, Lactobacillus plantarum.
Насыпным силосом обычно кормят молочный крупный рогатый скот, в то время как существует тенденция использования упакованного в тюки силоса для мясного крупного рогатого скота, овец и лошадей. Существует несколько преимуществ силоса в качестве корма для животных. В ходе ферментации, бактерии в силосе действуют на целлюлозу и другие углеводы в фураже с образованием органических жирных кислот, таким образом, понижая pH. Это подавляет конкурирующие бактерии, которые могут вызвать порчу, и органические кислоты, таким образом действуют в качестве естественных консервантов, улучшают перевариваемость и вкусовую привлекательность. Это действие в качестве консерванта является особенно важным в зимний период в регионах с умеренным климатом, когда зеленый фураж является недоступным.
Силос можно получать с использованием способов, известны в данной области, таких как описанные в CN 101940272 CN 103461658 CN 101946853, CN 101946853, CN 104381743, US3875304 и US 6224916. Гранулы для кормления животных можно получать с использованием способов, известных в данной области, таких как способы, описанные в US 3035920, US3573924 и US 5871802.
Биомасса растения
Повышение общего содержания липидов в биомассе растения эквивалентно более высокому содержанию энергии, что делает ее применение в качестве корма (продукта питания) или фуража или для продукции биологического топлива более экономичным.
Основным компонентом природной биомассы растения являются углеводы (приблизительно 75%, в пересчете на сухую массу) и лигнин (приблизительно 25%), который может варьировать в зависимости от вида растения. Углеводы в основном представляют собой целлюлозные или гемицеллюлозные волокна, которые придают прочность структуре растения, и лигнин, который удерживает волокна вместе. Биомассе растений обычно свойственна низкая плотность энергии в результате двух факторов: физической формы и содержания влаги. Это придает дополнительное неудобство и неэффективность ее хранению и транспортировке без какой-либо предварительной обработки.
Существует целый ряд доступных способов превращения ее в более пригодные формы, в том числе: 1) физическая предварительная обработка (например, размалывание), или 2) трансформация термическими (например, окисление, газификация, пиролиз) или химическими (например, анаэробное расщепление, ферментация, компостирование, переэтерификация) способами. Таким образом, биомассу превращают в то, что может быть описано, как топливная биомасса.
Сжигание
Сжигание представляет собой процесс, в ходе которого легковоспламеняющимся материалам позволяют гореть в присутствии воздуха или кислорода с высвобождением тепла. Основой процесса является окисление. Сжигание представляет собой самый простой способ использования биомассы для получения энергии и применим для обеспечения теплом. Указанное тепло может непосредственно использоваться множеством способов: 1) нагревание пространства, 2) нагревание воды (или другой жидкости) для центрального или местного нагревания или переработки тепла, 3) получение пара для генерации электричества или движущей силы. Если легковоспламеняющийся горючий материал находится в форме биомассы, окисление касается, в основном, атомов углерода (C) и водорода (H) в целлюлозе, гемицеллюлозе, лигнине и других присутствующих молекулах, с образованием диоксида углерода (CO2) и воды (H2O). Растения по изобретению дают усовершенствованное топливо для сжигания благодаря повышенному содержанию липидов.
Газификация
Газификация представляет собой процесс частичного окисления, посредством которого источник углерода, например, биомасса растения, разлагается на монооксид углерода (CO) и водород (H2), плюс диоксид углерода (CO2) и, возможно, молекулы углеводорода, например, метана (CH4). Если газификация имеет место при относительно низкой температуре, например, от 700°C до 1000°C, газообразный продукт будет содержать относительно высокие уровни углеводородов, по сравнению с высокотемпературной газификацией. В результате, он может использоваться непосредственно для сжигания с целью получения тепла или генерации электричества с помощью паровой турбины или, при условии соответствующего удаления газа, в двигателе внутреннего сгорания для генерации электричества. Камера сжигания для простого парового котла может быть соединена с газификатором, или устройство для получения газа может очищаться от углеводородов с более длинной цепью (смолы), которые хранятся и сжигаются в другом месте. Система газификации может быть тесно связана с газовой турбиной комбинированного цикла для генерации электричества (КЦИГ - комбинированный цикл интегрированной газификации). Высокотемпературная газификация (от 1200°C до 1600°C) приводит к образованию нескольких углеводородов в газообразном продукте и более высокой доле CO и H2. Это известно как синтетический газ (сингаз или биосинтетический газ), который может использоваться для синтеза углеводородов с более длинной цепью, с использованием таких способов, как синтез Фишера-Тропша (ФТ). При надлежащем соотношении H2 к CO (2:1), синтез ФТ может применяться для превращения синтетического газа в высококачественное синтетическое биологическое дизельное топливо, совместимое с обычными видами дизельного топлива из окаменелого сырья и дизельными двигателями.
Пиролиз
В настоящем описании термин «пиролиз» подразумевает процесс, в ходе которого используется медленное нагревание в отсутствие кислорода, с целью получения газообразных, нефтяных и угольных продуктов из биомассы. Пиролиз представляет собой термическое или термохимическое перевод материалов на основе липида, особенно на основе триглицерида. Продукты пиролиза включают газ, жидкость и твердый уголь, где доля каждого зависит от параметров процесса. Более низкие температуры (приблизительно 400°C) показывают тенденцию к образованию более твердого угля (медленный пиролиз), тогда как несколько более высокие температуры (приблизительно 500°C) дают намного более высокую долю жидкости (бионефть), при условии, что продолжительность обработки паров удерживается на уровне до приблизительно 1 секунды или менее.
Температура от приблизительно 275°C до приблизительно 375°C может применяться для получения жидкой бионефти с более высоким относительным содержанием углеводородов с большей длиной цепи. Пиролиз включает прямой термической крекинг липидов или комбинацию термического и каталитического крекинга. При температурах приблизительно 400-500°C происходит крекинг, дающий углеводороды с короткой цепью, например, алканы, алкены, алкадиены, ароматические вещества, олефины и карбоновую кислоту, а также монооксид углерода и диоксид углерода.
Могут использоваться четыре основных типа катализатора, в том числе, катализаторы на основе переходного металла, катализаторы типа молекулярного сита, активизированный алюминия оксид и натрия карбонат (Maher et al., 2007). Примеры представлены в US 4102938. Алюминия оксид (Al2O3), активированный кислотой, представляет собой эффективный катализатор (US 5233109). Катализаторы типа молекулярного сита представляют собой пористые, высоко кристаллические структуры, которые демонстрируют избирательность в отношении размера, таким образом, что сквозь них могут проникать только молекулы определенных размеров. Они включают катализаторы на основе цеолита, например, ZSM-5 или HZSM-5, которые являются кристаллическими материалами, содержащими AlO4 и SiO4, и другие катализаторы на основе кремния диоксида-алюминия оксида. Активность и селективность указанных катализаторов зависит от кислотности, размера пор и формы пор, и обычно они активны при 300-500°C. Катализаторы на основе переходного металла раскрыты, например, в US 4992605. Катализатор на основе натрия карбоната использовали в пиролизе масел (Dandik и Aksoy, 1998).
В настоящем описании «гидротермическая обработка», «ГТО», дополнительно называемая «термической деполимеризацией» представляет собой вид пиролиза, в ходе которого полученное из растения вещество, конкретно углеродсодержащий материал в полученном растение веществе, вводят в реакцию с водородом для получения продукта в форме бионефти, в основном состоящего из парафиновых углеводородов, наряду с другими газообразными и твердыми веществами. Значительное преимущество ГТО состоит в том, что нет необходимости сушить вегетативный растительный материал перед образованием композиции для введения в реакцию превращения, хотя вегетативный материал растения может быть предварительно высушен, чтобы упростить транспортировку или хранение биомассы. Биомасса может использоваться непосредственно после сбора урожая на поле. Реактор представляет собой любую емкость, способную выдерживать применяемую высокую температуру и давление и устойчивую к коррозии. Растворитель, применяемый для ГТО, содержит воду или полностью является водой или может содержать некоторые углеводородные соединения в форме масла. В общем, растворитель для ГТО не требует добавления спиртов. Реакция превращения может происходить в окислительном, восстановительном или инертном окружении. «Окислительный» в настоящем описании означает присутствие воздуха, «восстановительный» означает присутствие восстановительного агента, обычно газообразного водорода или метана, например, 10-15% H2 и остальная часть газообразного N2, и «инертный» означает присутствие инертного газа, такого как азот или аргон. Реакцию превращения предпочтительно, осуществляют в восстановительных условиях. Углеродсодержащие материалы для превращения включают целлюлозу, гемицеллюлозу, лигнин и белки, а также липиды. Для процесса применяют температуру превращения от 270°C до 400°C и давление от 70 до 350 бар, обычно от 300°C до 350°C и 100-170 бар. В результате процесса образуются пары органических веществ, пиролизные газы и древесный уголь. Пары органических веществ конденсируют для получения бионефти. Извлечение бионефти может быть осуществлено посредством охлаждения реактора и снижения давления до атмосферного, что позволяет образование бионефти (органическая) и водной фазы и выделение бионефти из реактора.
Выход выделенной бионефти вычисляют как процент от сухой массы исходной биомассы, в пересчете на сухую массу. Его вычисляют по формуле: масса бионефти x 100/сухая масса вегетативных частей растений. Масса бионефти не включает массу воды или твердых веществ, которые могут присутствовать в бионефтяной смеси и с легкостью удаляются фильтрацией или другими известными способами.
Далее бионефть можно разделить на фракции фракционной дистилляцией, с дополнительными процессами очистки или без них. Обычно, фракции, которые конденсируются при этих температурах, носят название: приблизительно 370°C, мазут; приблизительно 300°C, дизельное масло; приблизительно 200°C, керосин; приблизительно 150°C, газолин (бензин). Более тяжелые фракции можно расщеплять на более легкие, более желательные фракции, хорошо известные из уровня техники. Дизельное топливо обычно состоит из C13-C22 углеводородных соединений.
Переэтерификация
«Переэтерификация» в настоящем описании представляет собой перевод липидов, главным образом, триацилглицеринов, в метиловые эфиры или этиловые сложные эфиры жирных кислот посредством приведения в реакцию с короткоцепочечными спиртами, например, метанолом или этанолом, в присутствии катализатора, например, щелочи или кислоты. Метанол применим чаще за счет низкой стоимости и доступности, однако можно использовать также этанол, пропанол или бутанол или их смеси. Катализаторы могут быть гомогенными катализаторами, гетерогенными катализаторами или ферментными катализаторами. Гомогенные катализаторы включают сульфат железа, и затем KOH. Гетерогенные катализаторы включают CaO, K3PO4 и WO3/ZrO2. Ферментные катализаторы включают Novozyme 435, получаемый из Candida antarctica.
Переэтерификация может осуществляться на экстрагированном масле или предпочтительно, непосредственно in situ в вегетативном материале растения. Вегетативные части растения можно высушить и смолоть до использования, чтобы получить композицию для реакции превращения, но это не является необходимым. Преимущество прямого превращения в эфиры жирных кислот, предпочтительно, FAME, состоит в том, что для превращения можно применять более низкую температуру и давление, и несмотря на это получать высокий выход продукта, например, содержащего по меньшей мере 50% масс. FAME. Выход извлекаемой посредством переэтерификации бионефти вычисляют так же, как и для процесса ГТО.
Получение неполярных липидов
Способы, общепринятые в данной области, можно использовать применяться для выделения, обработки, очистки и анализа липидов, таких как TAG, продуцированных растениями или их частями по настоящему изобретению. Такие способы описаны и объяснены в источниках литературы, например, Fereidoon Shahidi, Current Protocols in Food Analytical Chemistry, John Wiley & Sons, Inc. (2001) D1.1.1-D1.1.11, и Perez-Vich et al. (1998).
Получение масла из вегетативных частей растений или семян
Обычно вегетативные части растений или семена растений варят, прессуют и/или экстрагируют для получения неочищенного растительного масла или масла из семян, которое в дальнейшем дегуммируют, рафинируют, отбеливают и дезодорируют. В общем, способы измельчения семени известны из уровня техники. Например, семена масличных культур можно размягчать обрызгиванием их водой, чтобы увеличить содержание влаги, например, до 8,5%, и вальцевать, используя гладкий валик со щелью 0,23-0,27 мм. В зависимости от вида семени, воду можно не добавлять до измельчения. Применение тепла инактивирует ферменты, облегчает дальнейшее разрушение клеток, слияние капелек липида и агломерацию частиц белка, каждое из которых облегчает процесс экстракции. Вегетативные части растений можно обрабатывать сходным образом, в зависимости от содержания влажности.
В одном варианте осуществления, большая часть растительного масла или масла из семян высвобождается при прохождении через винтовой пресс. Жмых (муку из вегетативных частей растений, жмыховую муку), выходящий из винтового пресса, можно в дальнейшем экстрагировать растворителем, например гексаном, с использованием колонки с подводом тепла, или можно не обрабатывать растворителем, в этом случае он может являться более подходящим в качестве пищевого продукта для животных. Альтернативно, неочищенное растительное масло или масло из семян, полученное способом прессования, может быть пропущено сквозь резервуар для отстаивания, оборудованный проволочным дренажным верхом, для удаления твердых веществ, которые сопровождают растительное масло или масло из семян в ходе операции прессования. Осветленное растительное масло или масло из семян может быть пропущено сквозь планшет и рамочный фильтр для удаления любых оставшихся тонких частиц твердого вещества.
Как только растворитель удаляют из неочищенного масла, прессованные и экстрагированные части объединяют и обрабатывают обычными способами обработки липидов (т.е., дегуммированием, рафинацией каустической содой, обесцвечиванием и дезодорированием).
Для экстракции липида из вегетативных частей растений по изобретению используют способы, аналогичные известным из уровня техники для экстракции масла из семян. Один из путей представляет собой физическую экстракцию, которая часто исключает экстракцию растворителем. Экстракция с помощью шнекового пресса, как и способы экстракции с помощью винтового пресса и гидравлического пресса, представляет собой распространенный вид экстракции. Механическая экстракция обычно менее эффективна, чем экстракция растворителем, при которой органический растворитель (например, гексан) смешивают по меньшей мере с биомассой растения, предпочтительно, после высушивания и перемалывания биомассы. Растворитель растворяет липид в биомассе и подобном материале, после чего раствор отделяют от биомассы механической операцией (например, с помощью описанных выше процессов прессования). Данная стадия отделения также может выполняться посредством фильтрации (например, с помощью пресс-фильтра или подобного устройства) или центрифугирования и т. п. В дальнейшем, органический растворитель может быть отделен от неполярного липида (например, дистилляцией). Вторая стадия разделения дает неполярный липид из растения и может давать повторно используемый растворитель, если он пригоден для традиционного сбора в виде паров. В одном варианте осуществления, содержание масла и/или белка в части растения или семени анализируют отражательной спектроскопией в ближней инфракрасной области, как описано в Hom et al. (2007), до экстракции.
Если вегетативные части растений не будут использовать для экстракции липида немедленно, их предпочтительно, перерабатывают, чтобы обеспечить максимальное сохранение содержания липида (см., например, Christie, 1993), например, посредством сушки вегетативных частей растений.
Дегуммирование
Дегуммирование является ранней стадией рафинирования масел, и его основная цель состоит в удалении из масла большинства фосфолипидов, которые могут присутствовать в количестве приблизительно 1-2% от общего экстрагированного липида. Добавление к неочищенному маслу ~2% воды, обычно содержащей фосфорную кислоту, при 70-80°C приводит к отделению большинства фосфолипидов, в сопровождении следовых количеств металлов и пигментов. Удаляемый нерастворимый материал, главным образом представляет собой смесь фосфолипидов и триацилглицеринов, и также известен как лецитин. Дегуммирование может осуществляться добавлением концентрированной фосфорной кислоты к неочищенному маслу, чтобы перевести неспособные к гидратации фосфатиды в способную к гидратации форму, и к незначительному количеству присутствующих хелатов металлов. Смолу отделяют от масла центрифугированием. Масло может быть рафинировано добавлением достаточного количества раствора натрия гидроксида для титрования всех жирных кислот, с удалением образованного таким образом мыла.
Щелочная рафинация
Щелочная рафинация представляет собой один из процессов очистки для обработки неочищенного масла, иногда также называемый нейтрализацией. Она обычно следует за дегуммированием и предшествует обесцвечиванию. После дегуммирования масло может быть обработано добавлением достаточного количества раствора щелочи, чтобы оттитровать все жирные кислоты и фосфорные кислоты, с последующим удалением образованного таким способом мыла. Пригодные щелочные материалы включают натрия гидроксид, калия гидроксид, натрия карбонат, лития гидроксид, кальция гидроксид, кальция карбонат и аммония гидроксид. Данный процесс обычно осуществляется при комнатной температуре и позволяет удалить фракцию свободных жирных кислот. Мыло удаляют центрифугированием или экстракцией растворителем мыла, и нейтрализованное масло промывают водой. При необходимости, любая дополнительная щелочь в масле может быть нейтрализована подходящей кислотой, например, хлористоводородной кислотой или серной кислотой.
Отбеливание
Отбеливание представляет собой процесс очистки, в ходе которого масла нагревают до 90-120°C, выдерживая при этой температуре в течение 10-30 минут в присутствии отбеливающей земли (0,2-2,0%) и в отсутствие кислорода, посредством проведения операции в атмосфере азота, под струей пара или в вакууме. Данная стадия обработки масла введена с целью удаления нежелательных пигментов (каротиноиды, хлорофилл, госсипол и т.д.), и в ходе данного процесса также удаляются продукты окисления, следовые металлы, соединения серы и следы мыла.
Дезодорирование
Дезодорирование представляет собой обработку масел и жиров при высокой температуре (200-260°C) и низком давлении (0,1-1 мм рт. ст). Это обычно достигается посредством введения пара в масло со скоростью приблизительно 0,1 мл/минуту/100 мл масла. Дезодорирование может выполняться посредством нагревания масла до 260°C под вакуумом, с медленным введением пара в масло со скоростью приблизительно 0,1 мл/минуту/100 мл масла. Приблизительно через 30 минут обрызгивания, маслу дают остыть под вакуумом. Масло обычно переносят в стеклянную емкость и продувают аргоном перед хранением при сниженной температуре. Если количество масла ограничено, масло может быть размещено под вакуумом, например, в реакторе Парра, и нагрето до 260°C с выдерживанием в течение такого периода времени, чтобы запах был уничтожен. Такая обработка улучшает цвет масла и удаляет большинство летучих веществ или пахучих соединений, включая любые оставшиеся свободные жирные кислоты, моноацилглицерины и продукты окисления.
Вымораживание
Вымораживание представляет собой процесс, иногда применяемый в коммерческом производстве масел для разделения масел и жиров на твердые (стеарин) и жидкие (олеин) фракции посредством кристаллизации при температурах ниже температуры окружающей среды. Впервые он был применен к хлопковому маслу с целью получения свободного от твердых веществ продукта. Его обычно применяют для уменьшения содержания насыщенных жирных кислот в маслах.
Водоросли
За год водоросли могут образовывать массу, в 10-100 раз превышающую массу наземных растений, где их можно выращивать в открытых водоемах (таких как водоемы по типу водоводов и озер) или в фотобиореакторах. Наиболее распространенные водоросли, вырабатывающие масло, в общем, могут включать диатомовые (бациллариофиты), зеленые водоросли (хлорофиты), сине-зеленые водоросли (цианофиты) и коричнево-золотистые водоросли (хризофиты). Дополнительно, пятая группа, известная как гаптофитовые водоросли, может быть использована. Группы включают коричневые водоросли и гетероконты. Конкретные неограничивающие примеры водорослей включают Классы: Chlorophyceae, Eustigmatophyceae, Prymnesiophyceae, Bacillariophyceae. Bacillariophytes, способные к продукции масла, включают рода Amphipleura, Amphora, Chaetoceros, Cyclotella, Cymbella, Fragilaria, Hantzschia, Navicula, Nitzschia, Phaeodactylum и Thalassiosira. Конкретные неограничивающие примеры хлорофитов, способных к продукции масла, включают Ankistrodesmus, Botryococcus, Chlorella, Chlorococcum, Dunaliella, Monoraphidium, Oocystis, Scenedesmus и Tetraselmis. В одном из аспектов, хлорофиты могут представлять собой Chlorella или Dunaliella. Конкретные неограничивающие примеры цианофитов, способных к продукции масла, включают Oscillatoria и Synechococcus. Конкретный пример хризофитов, способных к продукции масла, включает Boekelovia. Конкретные неограничивающие примеры гаптофитовых водорослей включают Isochysis и Pleurochysis
Конкретные водоросли, пригодные в соответствии с настоящим изобретением, включают, например, виды Chlamydomonas, например, Chlamydomonas reinhardtii, виды Dunaliella, например, Dunaliella salina, Dunaliella tertiolecta, D. acidophila, D. Lateralis, D. martima, D. parva, D. polmorpha, D. primolecta, D. pseudosalina, D. Quartolecta, D. viridis, виды Haematococcus, виды Chlorella, такие как Chlorella vulgaris, Chlorella sorokiniana или Chlorella protothecoides, виды Thraustochytrium, виды Schizochytrium, виды Volvox, виды Nannochloropsis, Botryococcus braunii, который может содержать более 60%, масс липида, Phaeodactylum tricornutum, Thalassiosira pseudonana, виды Isochrysis, виды Pavlova, виды Chlorococcum, виды Ellipsoidion, виды Neochloris, виды Scenedesmus.
Урожай водорослей по изобретению можно собирать с помощью микросит, центрифугирования, флоккуляции (с использованием, например, хитозана, алюминиевых квасцов и хлорида железа) и пенной флотации. Прерывание потока диоксида углерода может вынудить водоросли выпасть хлопьями самим по себе, что названа «аутофлоккуляцией». При пенной флотации, фермер аэрирует воду в пену, а затем снимает водоросли с поверхности. Ультразвуковой и другие способы сбора урожая в настоящий момент разрабатываются.
Липид может быть экстрагирован из водорослей механическим измельчением. При высушивании водорослевая масса сохраняет содержание липида, который в дальнейшем может быть «выжат» с помощью масляного пресса. Кроме того, осмотический шок может быть применен для высвобождения клеточных компонентов, например, липида из водорослей, а выделение с помощью ультразвука может ускорить процессы экстракции. Химические растворители (например, гексан, бензол, петролейный эфир) часто применяются для экстракции липидов из водорослей. Дополнительно, ферментная экстракция с использованием ферментов для разложения клеточных стенок может применяться для выделения липидов из водорослей. Кроме того, суперкритический CO2 может использоваться в качестве растворителя. В данном способе CO2 превращают в жидкость под давлением и нагревают до точки, в которой он становится суперкритическим (обладает свойствами как жидкости, так и газа), что дает ему возможность выполнять роль растворителя.
Применение растительных липидов
Липиды, полученные описанными способами, находят множество применений. В некоторых вариантах осуществления липиды используются в качестве пищевых масел. В других вариантах осуществления липиды рафинируют и используют в качестве смазки или для других промышленных целей, например, синтеза пластмасс. В некоторых предпочтительных вариантах осуществления липиды рафинируют для производства биодизельного топлива. Биодизельное топливо может быть получено из масел, получаемых из растений, водорослей и грибов по изобретению. Применение растительных триацилглицеринов для получения биологического топлива рассмотрено в Durrett et al. (2008). Полученное топливо обычно обозначается как биодизельное топливо и обладает динамическим интервалом вязкости 1,9-6,0 мм2с-1 (ASTM D6751). Биоспирт можно получить из сахаров биомассы, не принадлежащих к липидам, которые остаются после экстракции липида. Общие способы получения биологического топлива могут быть обнаружены, например, в Maher и Bressler, 2007; Greenwell et al., 2010; Karmakar et al., 2010; Alonso et al., 2010; Liu et al., 2010a. Gong и Jiang, 2011; Endalew et al., 2011; и Semwal et al., 2011.
В настоящем изобретении предлагаются способы повышения содержания масла в вегетативных тканях. Растения по настоящему изобретению содержат повышенное количество энергии в листьях и/или стеблях, таким образом, что цельные надземные части растения можно собирать и использовать для получения биотоплива. Кроме того, уровень олеиновой кислоты значительно повышен, притом, что уровень полиненасыщенной жирной альфа-линоленовой кислоты (ALA) снижен. Таким образом, растения, морские водоросли и грибы по настоящему изобретению снижают затраты на производство биотоплива.
Биодизельное топливо
Производство биодизельного топлива или алкиловых сложных эфиров хорошо известно. Существует три основных способа получения сложного эфира из липидов: 1) катализированная основанием переэтерификация липида с использованием спирта; 2) прсилосная ямая, катализируемая кислотой этерификация липида с использованием метанола; и 3) перевод липида в жирные кислоты, а затем в алкиловые эфиры с помощью кислотного катализа Может применяться любой способ получения алкиловых сложных эфиров и глицериловых эфиров жирных кислот (в ходе которого этерифицируются одна, две или три гидроксигруппы на глицерине). Жирные кислоты могут быть получены, например, гидролизом или омылением TAG с использованием кислотных или основных катализаторов, соответственно, или фермента, например, липазы или эстеразы. Алкиловые эфиры жирных кислот могут быть получены посредством введения жирной кислоты в реакцию со спиртом в присутствии кислотного катализатора. Кроме того, алкиловые эфиры жирных кислот могут быть получены посредством введения TAG в реакцию со спиртом в присутствии кислотного или основного катализатора.
Эфиры глицерина могут быть получены, например, введением глицерина в реакцию с алкилгалогенидом в присутствии основания или с олефином или спиртом в присутствии кислотного катализатора. Алкиловые эфиры могут быть непосредственно смешаны с дизельным топливом или промыты водой или другими водными растворами с целью удаления различных примесей, в том числе катализаторов, перед смешиванием.
Авиационное топливо
Для улучшения характеристик биотоплива разработаны технологии термического и каталитического расщепления (разрыва) химических связей, что позволяет превращать бионефть в биологические альтернативы полученному из нефть дизельному топливу и другим видам топлива, такие как реактивное топливо.
Применение источника среднецепочечных жирных кислот, такого как образуемый клеткой по изобретению, растением или его частью по изобретению, семенем по изобретению или трансгенным вариантом любого из них, предотвращает потребность в высоких энергиях для расщепления цепи жирной кислоты и получения более коротких молекул, необходимых для реактивного топлива и других видов топлива с требованиями низкой температуры потока. Этот способ включает отщепление одной или более групп среднецепочечных жирных кислотных от глицеридов с получением глицерина и одной или нескольких свободных жирных кислот. Кроме того, способ включает отщепление одной или нескольких среднецепочечных жирных кислот от глицерина и декарбоксилирование одной или несккольких среднецепочечных жирных кислот с получением одного или нескольких углеводородов для получения реактивного топлива.
Составы
Настоящее изобретение также относится к составам, особенно, фармацевтическим составам, содержащим одно или несколько из растений, частей растений, липидов, белков, содержащих азот молекул или содержащих углерод молекул полученных с использованием способов по изобретению.
Фармацевтический состав может дополнительно содержать активный ингредиент и стандартный, известный, нетоксичный фармацевтически приемлемый носитель, адъювант или растворитель, такой как фосфатно-солевой буфер, вода, этанол, полиолы, растительные масла, увлажнитель или эмульсия, например, эмульсия вода/масло. Состав может находиться в жидкой или твердой форме. Например, состав может находиться в форме таблетки, капсулы, жидкости для приема внутрь, порошка, мази или крема для местного применения. Надлежащая сыпучесть может поддерживаться, например, поддержанием необходимого размера частиц в случае дисперсий и применением поверхностно-активных веществ. Кроме того, может быть желательным введение поддерживающих изотоничность агентов, например, сахаров, натрия хлорида и т. п. Помимо таких инертных разбавителей, состав может включать вспомогательные вещества, например, увлажнители, эмульгаторы и суспендирующие агенты, подсластители, вкусовые добавки и ароматизаторы.
Типичные дозы конкретной жирной кислоты составляют от 0,1 мг до 20 г, для приема от 1 до 5 раз в сутки (до 100 г в сутки) и, предпочтительно, находятся в интервале от приблизительно 10 мг до приблизительно 1, 2, 5 или 10 г в сутки (в виде одной или нескольких доз). Как известно в данной области, желательной является доза по меньшей мере приблизительно 300 мг/сутки жирной кислоты, особенно полиненасыщенной жирной кислоты. Однако следует понимать, что любое количество жирной кислоты будет полезным для субъекта.
Возможные способы введения фармацевтических составов по настоящему изобретению включают, например, энтеральный и парентеральный. Например, жидкий препарат можно вводить перорально. Дополнительно, гомогенная смесь может быть полностью диспергирована в воде, смешана в стерильных условиях с физиологически приемлемыми разбавителями, консервантами, буферами или пропеллентами, чтобы получить спрей или средство для ингаляций.
Специалист в данной области может определять дозы состава для введения субъекту, и они зависят от различных факторов, таких как масса тела, возраст, общее состояние здоровья, анамнез, иммунный статус субъекта и т. п.
Дополнительно, составы по настоящему изобретению можно использовать для косметических целей. Составы можно добавлять к уже существующим косметическим составам, таким образом, что образуется смесь, или жирную кислоту, полученную по изобретению, можно использовать в качестве единственного «активного» ингредиента в косметическом составе.
Полипептиды
В настоящем описании термины «полипептид» и «белок» в общем используют взаимозаменяемо.
Полипептид или класс полипептидов можно определять по степени идентичности (% идентичности) его последовательности аминокислот с эталонной последовательностью аминокислот или большему % идентичности с одной эталонной последовательностью аминокислот, чем с другой. % идентичности полипептида с эталонной последовательность аминокислот обычно определяют посредством анализа GAP (Needleman and Wunsch, 1970; программное обеспечение GCG) с параметрами штрафа за открытие пропуска=5, и штрафа на продление пропуска=0,3. Длина последовательности запроса составляет по меньшей мере 100 аминокислот, и анализ GAP выравнивает две последовательности на протяжении участка длиной по меньшей мере 100 аминокислот. Даже более предпочтительно, длина последовательности запроса составляет по меньшей мере 250 аминокислот, и анализ GAP выравнивает две последовательности на протяжении участка длиной по меньшей мере 250 аминокислот. Даже более предпочтительно, анализ GAP выравнивает две последовательности на протяжении их полной длины, и степень идентичности определяют на протяжении полной длины эталонной последовательности. Полипептид или класс полипептидов может обладать такой же или отличной ферментной активностью, по сравнению с эталонным полипептидом, или может не обладать активностью эталонного полипептида. Предпочтительно, полипептид обладает ферментной активностью, составляющей по меньшей мере 10% от активности эталонного полипептида.
В настоящем описании «биологически активный фрагмент» представляет собой часть полипептида по изобретению, которая обладает определенным видом активности полноразмерного эталонного полипептида, например, активностью DGAT. Биологически активные фрагменты в настоящем описании исключают полноразмерный полипептид. Биологически активные фрагменты могут быть частью любого размера, до тех пор, пока они сохраняют определенную активность. Предпочтительно, биологически активный фрагмент сохраняет по меньшей мере 10% активности полноразмерного полипептида.
Относительно определенного полипептида или фермента, предусматривается, что более высокий % идентичности, чем раскрытый в настоящем описании, будет включать предпочтительные варианты. Таким образом, если это уместно, в свете минимального % идентичности, предпочтительно, полипептид/фермент содержит последовательность аминокислот, которая по меньшей мере на 60%, более предпочтительно, по меньшей мере на 65%, более предпочтительно, по меньшей мере на 70%, более предпочтительно, по меньшей мере на 75%, более предпочтительно, по меньшей мере на 80%, более предпочтительно, по меньшей мере на 85%, более предпочтительно, по меньшей мере на 90%, более предпочтительно, по меньшей мере на 91%, более предпочтительно, по меньшей мере на 92%, более предпочтительно, по меньшей мере на 93%, более предпочтительно, по меньшей мере на 94%, более предпочтительно, по меньшей мере на 95%, более предпочтительно, по меньшей мере на 96%, более предпочтительно, по меньшей мере на 97%, более предпочтительно, по меньшей мере на 98%, более предпочтительно, по меньшей мере на 99%, более предпочтительно, по меньшей мере на 99,1%, более предпочтительно, по меньшей мере на 99,2%, более предпочтительно, по меньшей мере на 99,3%, более предпочтительно, по меньшей мере на 99,4%, более предпочтительно, по меньшей мере на 99,5%, более предпочтительно, по меньшей мере на 99,6%, более предпочтительно, по меньшей мере на 99,7%, более предпочтительно, по меньшей мере на 99,8%, и даже более предпочтительно, по меньшей мере на 99,9% идентична соответствующей указанной SEQ ID NO.
Мутантные последовательности аминокислот полипептидов, раскрытых в настоящем описании, могут быть получены посредством введения подходящих нуклеотидных модификаций в нуклеиновую кислоту, определенную в настоящем описании, или синтезом желательного полипептида in vitro. Такие мутанты включают, например, делеции, вставки или замены остатков в пределах последовательности аминокислот. Комбинация делеций, вставок и замен может быть получена для введения в конечную конструкцию, при условии, что конечный полипептидный продукт обладает желательными характеристиками.
Мутантные (модифицированные) полипептиды могут быть получены с использованием любой техники, известной в данной области, например, с использованием стратегий направленной эволюции или рационального дизайна (см. ниже). Для продуктов, получаемых из мутантной/модифицированной ДНК, может быть легко проведен скрининг с использованием способов, раскрытых в настоящем описании, чтобы определить, обладают ли они активностью фактора транскрипции, ацилтрансферазы жирных кислот или OBC.
При конструировании мутантов последовательности аминокислот, расположение сайта мутации и природа мутации будут зависеть от модифицированной характеристики (характеристик). Сайты мутации могут быть модифицированы индивидуально или сериями, например, (1) сначала заменой консервативными альтернативами аминокислот, а затем более радикальными вариантами, в зависимости от полученных результатов, (2) удалением целевого остатка, или (3) вставкой других остатков, смежных с размещенным сайтом.
Длина делеций в последовательности аминокислот, в общем, варьирует от приблизительно 1 до 15 остатков, более предпочтительно, приблизительно 1-10 остатков и обычно приблизительно 1-5 смежных остатков.
В мутантах с заменой по меньшей мере один остаток аминокислоты в полипептиде удален, и другой остаток вставлен на его место. Сайты, представляющие наибольший интерес для мутагенеза с замещением с целью инактивации ферментов, включают сайты, идентифицированные как активный(е) сайт(ы). Другими сайтами, представляющими интерес, являются такие, конкретные остатки которых, полученные от различных штаммов или видов, идентичны. Указанные положения могут быть важны с точки зрения биологической активности. Такие сайты, особенно находящиеся в пределах последовательности по меньшей мере трех других одинаково консервативных сайтов, предпочтительно, заменяются относительно консервативным образом. Такие консервативные замены приведены в Таблице 1 под заголовком «примеры замен».
Таблица 1. Примеры замен.
В предпочтительном варианте мутантный/вариантный полипептид содержит только или не более чем одну, две, три или четыре консервативных модификации аминокислот, по сравнению с природным полипептидом. Подробности относительно консервативных модификаций аминокислот приведены в Таблице 1. Как понятно специалисту в данной области, такие незначительные модификации могут быть спрогнозированы на рациональной основе как не влияющие на активность полипептида при экспрессии в трансгенном растении или его части. Мутанты с желательной активностью можно конструировать с использованием стандартных в данной области способов, например, случайным мутагенезом, направленным мутагенезом или мутагенезом насыщения в известных целевых генах, или посредством обработки различных генов «перетасовкой» ДНК.
ПРИМЕРЫ
Пример 1. Общие материалы и способы
Экспрессия генов в клетках растений в системе временной экспрессии
Гены экспрессируют в клетках растения с использованием системы временной экспрессии, по существу как описано в Voinnet et al. (2003) и Wood et al. (2009). Бинарные векторы, содержащие кодирующий участок для экспрессии посредством высокоактивного конститутивного промотора e35S, содержащего дуплицированный энхансерный участок, вводят в Agrobacterium tumefaciens штамм AGL1. Химерный бинарный вектор, 35S:p19, для экспрессии вирусного супрессора сайленсинга p19 вводят в AGL1 отдельно, как описано в WO2010/057246. Химерный бинарный вектор, 35S:V2, для экспрессии вирусного супрессора сайленсинга V2 вводят в AGL1 отдельно. Рекомбинантные клетки выращивают до стационарной фазы при температуре 28°C в среде Лурия-Бертани с добавлением 50 мг/л канамицина и 50 мг/л рифампицина. Затем бактерии осаждают центрифугированием при 5000 g в течение 5 минут при комнатной температуре, перед ресуспендированием до OD600=1,0 в инфильтрационном буфере, содержащем 10 мМ минимальной эссенциальной среды, pH 5,7, 10 мМ MgCl2 и 100 мкМ ацетосирингона. Далее клетки инкубируют при 28°C со встряхиванием в течение 3 часов, после чего измеряют OD600, и объем каждой культуры, содержащей конструкцию вирусного супрессора 35S:p19 или 35S:V2, необходимый для достижения конечной концентрации OD600=0,125, помещают в чистую пробирку. Доводят до конечного объема упомянутым выше буфером. После этого осуществляют инфильтрацию листьев смесью культуры, и растения обычно выращивают еще в течение 3-5 суток после инфильтрации, перед выделением листовых пластинок для получения очищенного лизата клеток или выделения общего липида.
Трансформация Sorghum bicolor L.
Растительный материал
Растения сорго из инбредного культивара TX-430 (Miller, 1984) выращивали в камере для выращивания растений (Conviron, PGC-20 flex) при «дневной» температуре 28±1°C и «ночной» температуре 20±1°C, с фотопериодом 16 час с интенсивностью света в течение «дня» 900-1000 люкс. Метелки накрывали полупрозрачными бумажными пакетами до цветения. Незрелые зародыши собирали из метелок через 12-15 суток после цветения. Метелки промывали несколько раз водой, и развившиеся семена, которые были равномерными по размеру, выделяли и подвергали поверхностной стерилизации с использованием 20% коммерческого отбеливателя, смешанного с 0,1% Tween-20, в течение 15-20 мин. Затем их промывали стерильной дистиллированной водой 3 раза по 20 мин каждый, и промокали досуха в ламинарном шкафу. Незрелые зародыши (IE) длиной в диапазоне от 1,4 до 2,5 мм асептически выделяли в ламинарном шкафу и использовали в качестве исходной ткани для получения зеленой регенерирующей ткани.
Базовые среды для культивирования
Среды, используемые для трансформации растений, были основаны на MS (Murashige and Skoog, 1962), поставляемой из PhytoTechnology Laboratories (M519). pH сред доводили до 5,8 перед стерилизацией при 121°C в течение 15 мин. Термочувствительные регуляторы роста растений и другие добавки, такие как генетицин (G418, Sigma), используемый в качестве селективного средства, стерилизовали фильтрацией (0,2 мкм) и добавляли в среду после стерилизации, когда среду охлаждали до приблизительно 55°C. Оптимизированный состав культуральной среды для различных стадий трансформации растений от индукции образования каллюса до регенерации растения из зеленой ткани, индуцированной из незрелых зародышей, представлены в Таблице 2.
Способы и материалы для культивирования
Выделенные IE длиной в диапазоне от 1,4 до 2,5 мм помещали в среды для индукции образования каллюса-осмотическую среду (CIM-осмотическую среду, Таблица 2) с щитком спереди. Базовую среду CIM модифицировали для улучшения качества каллюса и частоты индукции из незрелых зародышей, так же как среду для регенерации каллюса, посредством включения α-липоевой кислоты (1-5 мг/л), мелатонина (5-10 мг/л) и 2-аминоиндан-2-фосфоновой кислоты HCl (1-2 мг/л), если не указано иначе. Для развития зеленой ткани, незрелые зародыши инкубировали под флуоресцентным светом 45-50 мколь с-1 м-2 (16 час/сутки) в помещении для культивирования клеток при 24±2°C. Через трое суток культивирования, корень и полюсы побегов незрелых зародышей асептически отделяли и повторно инокулировали в такую же CIM и поддерживали в таких же условиях, как описано выше. Их субкультивировали каждые две недели в такой же CIM в течение 6 недель и оценивали по качеству каллюса, эффективности индукции образования каллюса и эффективности трансформации.
Каллюсы, образование которых было инициировано из IE в первые 3-4 недели на CIM, являлись по большей части эмбриогенными и медленно подвергались дифференцировке в эмбриогенный каллюс с узловатыми структурами с окраской от светло- до более темно-зеленого. Эмбриогенные каллюсы с зелеными узловатыми структурами отбирали и поддерживали в такой же среде (CIM) посредством субкультивирования каждые 2 недели в течение вплоть до 6 месяцев или более, для использования в качестве эксплантатов для трансформации. Этот тип ткани назван в настоящем описании как ткань «дифференцирующегося эмбриогенного каллюса» или «DEC», поскольку эта ткань формирует узловатые структуры из дифференцирующихся клеток, сохраняющие эмбриогенный и органогенный потенциал, даже несмотря на то, что ткани фактически представляли собой смесь клеток каллюса, клеток, формирующих узловатые структуры и гранулярные структуры, и промежуточных клеток, которые авторы настоящего изобретения считали находящимися на пути развития где-то между каллюсом (представляющим собой недифференцированные клетки) и узловатыми структурами. Иногда ткани включали соматические зародыши на ранних стадиях (глобулярные).
Таблица 2. Среды, использованные в индукции образования ткани DEC и трансформации сорго
Бомбардировка частицами зеленых регенерирующих тканей DEC
Плазмиды, содержащие ген селективного маркера, кодирующий неомицин-фосфотрансферазу II (NptII), обеспечивающую устойчивость к антибиотику генетицину, под контролем промотора pUbi и с терминацией посредством 3'-области nos, конструировали или получали для экспериментов для получения стабильной трансформации или для совместной бомбардировки с другими плазмидами. Плазмидные ДНК выделяли с использованием набора Zymopure™ Maxiprep (USA) в соответствии с инструкциями производителя. В качестве контрольного вектора для трансформации, получали генетический вектор, содержащий гены uidA (GUS) и bar, сконструированные для экспрессии в клетках растений. Ген uidA помещали под регуляторный контроль промотора полиубиквитина (pUbi) маиса и последовательности полиаденилирования/терминатора октопинсинтазы (ocs 3') Agrobacterium tumefaciens. Последовательность между промотором и кодирующей областью белка включала 5' UTR и первый интрон гена Ubi. Присутствовал также репортерный ген uidA, внутри кодирующей области белка, интрон из гена каталазы клещевины, предотвращающий трансляцию функционального белка GUS в Agrobacterium, таким образом, уменьшая фоновую экспрессию гена GUS в инокулированных тканях растений. Таким образом, любая экспрессия GUS может быть обусловлена экспрессией гена uidA в клетках растений. Ген bar также помещали под регуляторный контроль промотора pUbi и терминировали с использованием 3'-регуляторной последовательности нопалинсинтазы (nos 3') Agrobacterium. Вектор uidA/bar сначала использовали в экспериментах для детекции временной экспрессии гена в тканях DEC сорго.
Равномерно здоровые, зеленые регенерирующие ткани DEC (размером 4-5 мм), полученные с использованием способов, описанных выше, и культивированные в течение от 6 недель до 6 месяцев от начала, использовали для опосредованной микрочастицами трансформации (бомбардировки) с использованием плазмид. Приблизительно 15 равномерно зеленых тканей DEC (4-5 мм каждая) помещали в центр чашки Петри (диаметром 90 мм), содержащей CIM - осмотическую среду (Таблица 2), и инкубировали в темноте в течение приблизительно 4 час до бомбардировки. Бомбардировку проводили с использованием устройства PDS-1000 He (Biorad, Hercules, CA), как описано в Liu et al. (2014). После бомбардировки, ткани поддерживали в той же самой осмотической среде в течение ночи и переносили в среду для предварительного отбора на следующее утро
Зеленые ткани DEC, бомбардированные с использованием генетической векторной плазмиды, имеющей селективный маркер, кодирующий NptII, переносили в среду CIM-PS на 3-4 суток до какого-либо отбора, с добавлением в среду двух соединений в качестве антиоксидантов, L-цистеина (50 мг/л) и аскорбиновой кислоты (15 мг/л) (Таблица 2). Без добавления этих антиоксидантов в среду для предварительного отбора, многие из бомбардированных тканей приобретали коричневую, некоторые достаточно темную коричневую окраску, и многие теряли любую способность к дальнейшему росту. После 3-4 суток в среде для предварительного отбора, некоторые из бомбардированных тканей подвергали окрашиванию GUS и рассматривали под микроскопом для подсчета отдельных синих (положительных по GUS) пятен, для проверки того, что гены перенесены и могут быть экспрессированы. Включение двух антиоксидантов в среде для предварительного отбора улучшало эффективность трансформации, как показано по временной экспрессии гена GUS.
Отбор и регенерация трансгенных растений в оптимизированных условиях
После бомбардировки и 3-4 суток культивирования в среде для предварительного отбора без селективного средства (генетицина), размер бомбардированных тканей увеличился от 4-5 мм до приблизительно 6-7 мм. Эти ткани переносили в селективную среду CIM/G25, содержащую 25 мг/л генетицина (Таблица 2) и культивировали в течение дополнительных 4 недель. Когда возможно, бомбардированные ткани разделяли на 2-6 фрагментов каждая, увеличивая выделение независимых трансформантов. Все ткани культивировали в среде, как описано в Таблице 2 и поддерживали для регенерации предположительных трансгенных растений.
Растения эффективно регенерировали после роста на этих средах. Каждую бомбардированную ткань и побеги, полученные из нее, субкультивировали и поддерживали отдельно для расчета эффективности трансформации. Положительную трансформацию подтверждали посредством ПЦР на геномной ДНК растения, выделенной из образцов побегов, показывающий присутствие гена селективного маркера. Количество трансформантов рассчитывали в пересчете на ввод ткани DEC. Получали эффективность трансформации приблизительно 50%, выраженную как количество независимых трансформантов на ввод бомбардированной ткани.
Опосредованная Agrobacterium трансформация зеленых регенерирующих тканей DEC
Равномерно здоровые, зеленые регенерирующие ткани DEC (размером 4-5 мм), полученные с использованием способов, описанных в предшествующих примерах, и культивированные в течение от 6 недель до 6 месяцев от начала, использовали для опосредованной Agrobacterium трансформации.
Генетические векторы, имеющие области T-ДНК, содержащие гены для трансформации, конструировали и получали для трансформации зеленых регенерирующих тканей DEC с использованием опосредованной Agrobacterium трансформации. Контрольный бинарный вектор, содержащий гены uidA (GUS) и bar, конструировали для экспрессии в клетках растений. Ген uidA gene помещали под регуляторный контроль промотора полиубиквитина (pUbi) маиса и последовательности полиаденилирования/терминатора октопинсинтазы (ocs 3') Agrobacterium tumefaciens. Последовательность между промотором и кодирующей областью белка включала 5' UTR и первый интрон гена Ubi. Присутствовал также репортерный ген uidA, внутри кодирующей области белка, интрон из гена каталазы клещевины, предотвращающий трансляцию функционального белка GUS в Agrobacterium, таким образом, уменьшая фоновую экспрессию гена GUS в инокулированных тканях растений. Таким образом, любая экспрессия GUS была обусловлена экспрессией гена uidA в клетках растений. Ген bar также помещали под регуляторный контроль промотора pUbi и терминировали с использованием 3'-регуляторной последовательности нопалинсинтазы (nos 3') Agrobacterium нопалинсинтазы (nos 3').
Получали пригодный штамм Agrobacterium tumefaciens, например, AGL1, как описано в Lazo et al. (1991), и генетический вектор вводили в штамм Agrobacterium tumefaciens способом теплового шока.
Культуры Agrobacterium, несущие генетическую конструкцию, выращивают в пригодной среде, например, среде LB, и в подходящих условиях для получения инокулята Agrobacterium, после чего равномерно здоровые, зеленые регенерирующие ткани DEC инфицируют инокулятом Agrobacterium. Инфицированные ткани DEC промокают стерильной фильтровальной бумагой для удаления избытка Agrobacterium и переносят в среду для совместного культивирования, необязательно, дополненную антиоксидантами, и инкубируют в темноте при приблизительно 22-24°C в течение 2-4 суток. После инкубации, ткани DEC обрабатывают подходящим средством для уничтожения Agrobacterium, промывают в стерильной воде, переносят на подходящую среду и позволяют расти. Через 4-6 недели, побеги вырезают и культивируют на среде для удлинения побегов, после чего предположительно трансгенные побеги детектируют с использованием подходящих анализов.
Трансформация Brassica napus
Семена Brassica napus стерилизуют с использованием газообразного хлора, как описано Kereszt et al. (2007), и проращивают на питательной среде для культуры тканей. Котиледонные черешки с ножкой 2-4 мм выделяют, как описано Belide et al. (2013) и используют в качестве эксплантов. Готовят культуры A. tumefaciens AGL1 (Lazo et al., 1991), содержащие бинарный вектор, и котиледонные черешки инокулируют культурами, как описано Belide et al. (2013). Инфицированные котиледонные черешки культивируют на среде МС с добавлением 1 мг/л тидиазурона (ТДЗ)+0.1 мг/л 1-нафталинуксусной кислоты (НУК)+3 мг/л AgNO3+250 мг/л цефотаксима, 50 мг/л тиментина и 25 мг/л канамицина и культивируют в течение 4 недель при 24°C с фотопериодом света-темноты 16 часов/8 часов, с двухнедельными пассажами на такую же среду. Экспланты с зеленым каллюсом переносят в среду для инициации побегов (МС+1 мг/л кинетина+3 мг/л AgNO3+250 мг/л цефотаксима+50 мг/л тиментина+25 мг/л канамицина) и культивируют еще в течение 2-3 недель. Маленькие побеги (~1 см) выделяют из устойчивого каллюса и переносят в среду для удлинения побегов (среда МС с добавлением 0,1 мг/л гибберелловой кислоты+3 мг/л AgNO3+250 мг/л цефотаксима+25 мг/л канамицина) и культивируют еще в течение двух недель. Здоровые побеги с одним или двумя листьями отбирают и переносят в среду для корнеобразования (1/2 МС с добавлением 1 мг/л НУК+20 мг/л аденина сульфата (АДС)+3 мг/л AgNO3+250 мг/л цефотаксима) и культивируют в течение 2-3 недель. ДНК выделяют из маленьких листьев устойчивых побегов с использованием набора для выделения ДНК растения (Bioline, Alexandria, NSW, Australia), в соответствии с протоколом производителя. Присутствие последовательностей T-ДНК определяют способом ПЦР амплификации на геномной ДНК. Положительные, трансгенные побеги с корнями переносят в горшки, содержащие смесь для выращивания саженцев и выращивают в теплице с дневной температурой 24°C/ночной температурой 16°C (стандартные условия).
Анализ ферментов очищенного лизата листа
Ткани листа Nicotiana benthamiana, предварительно обработанные инфильтрацией, как описано выше, измельчают в растворе, содержащем 0,1 М калий фосфатного буфера (pH 7,2) и 0,33 М сахарозы, с использованием стеклянного гомогенизатора. Гомогенат листьев центрифугируют при 20 000 g в течение 45 минут при температуре 4°C, после чего собирают каждый супернатант. Содержание белка в каждом супернатанте измеряют согласно Bradford (1976), с помощью мультиметочного счетчика Wallac1420 и реактива красителя для анализа белка Bio-Rad (Bio-Rad Laboratories, Hercules, CA USA). В анализах ацилтрансферазы используют 100 мкг белка согласно Cao et al. (2007), с некоторыми модификациями. Реакционная среда содержит 100 мМ Трис-HCl (pH 7,0), 5 мМ MgCl2, 1 мг/мл альбумина телячьей сыворотки (не содержащего жирных кислот), 200 мМ сахарозы, 40 мМ холодного олеоил-CoA, 16,4 мкМ sn-2 моноолеоилглицерил[14C] (55 мКи/ммоль, American Radiochemicals, Saint Louis, MO USA) или 6,0 мкМ динатриевой соли [14C]глицерил-3-фосфата (G-3-P) (150 мКи/ммоль, American Radiochemicals). Анализ проводят через 7,5, 15 или 30 минут.
Анализ липидов
Анализ содержания масла в семенах
Если содержание масла в семени или состав общих жирных кислот следует определить для семян небольшого размера, например, семян Arabidopsis, то жирные кислоты в семенах метилируют напрямую, без раздавливания семян. Семена сушат в эксикаторе в течение 24 часов, и приблизительно 4 мг семян переносят в стеклянный флакон емкостью 2 мл с навинчивающейся крышкой, покрытой слоем Тефлона. Раствор 0,05 мг тригептадеканоина (TAG с тремя C17:0 жирными кислотами) в 0,1 мл толуола помещают во флакон в качестве внутреннего стандарта. Жирные кислоты семени метилируют добавлением 0,7 мл 1 н метанольного HCl (Supelco) во флакон, содержащий материал семян. В дроблении семян для полного метилирования нет необходимости в случае семян небольшого размера, например, семян Arabidopsis. Смесь кратковременно обрабатывают вихревым перемешиванием и инкубируют при 80°C в течение 2 часов. После охлаждения смесей до комнатной температуры, 0,3 мл 0,9% NaCl (масс/об) и 0,1 мл гексана добавляют во флакон и тщательно перемешивают в течение 10 минут в Heidolph Vibramax 110. FAME собирают в стеклянную вставку емкостью 0,3 мл и анализируют способом GC с плазменно-ионизационным детектором (ПИД), как описано ниже.
Площадь пика индивидуальных FAME сначала корректируют на основе площади пиков известного количества таких же FAME, присутствующих в коммерческом стандартном GLC-411 (NU-CHEK PREP, INC., США). GLC-411 содержит равные количества 31 жирной кислоты (% масс.), в интервале от C8:0 до C22:6. В случае жирных кислот, которые не присутствовали в стандарте, находят площадь пиков наиболее подобного FAME. Например, площадь пика FAME 16:1d9 используют для 16:1d7, и площадь пика FAME C22:6 используют для C22:5. Скорректированные площади пиков используют для вычисления массы каждого FAME в образце, по сравнению с массой внутреннего стандарта. Масло хранится, в основном, в форме TAG, и его массу вычисляют на основании массы FAME. Общее количество моль глицерина определяют посредством вычисления количества моль каждого FAME и деления общего количества моль FAME на 3. Содержание TAG вычисляют как сумму фрагментов глицерина и жирного ацила, с использованием формулы: %, масс. масла=100 x ((41 x общее количество моль FAME/3)+(общее количество г FAME - (15 x общее количество моль FAME)))/грамм семени, где 41 и 15 представляют собой значения молекулярной массы фрагмента глицерина и метильной группы, соответственно.
Анализ содержания жирной кислоты в семенах большего размера
Для определения состава жирных кислот в одиночных семенах большего размера, таких как семена рапса и Camelina, или семена Sorghum или кукурузы, выполняют прямое метилирование жирных кислот в семени, как и для семян Arabidopsis, за исключением разрушения оболочек семян. Данный способ позволяет экстрагировать достаточное количество масла из семян, чтобы провести анализ состава жирных кислот. Для определения состава жирных кислот общего липида, экстрагированного из семян, семена раздавливают и липиды экстрагируют с помощью CHCl3/MeOH. Аликвоты экстрагированного липида метилируют и анализируют способом GC. Содержание общего липида для объединенного в пул семени рапса (содержание масла в семенах) определяют при помощи двукратной экстракции липида с использованием CHCl3/MeOH из известной массы высушенных семян после раздавливания, с последующим метилированием аликвот липида вместе с 17:0 жирными кислотами в качестве внутреннего стандарта. В случае семян большего размера, таких как Camelina, липид из известного количества семян метилируют вместе с известным количеством 17:0 жирных кислот, как и в случае анализа масла Arabidopsis, и FAME анализируют способом GC. Для количественного определения TAG, фракционируют TAG из экстрагированного липида с использованием TLC и непосредственно метилируют на кремния диоксиде с использованием 17:0 TAG в качестве внутреннего стандарта. Эти способы описаны более подробно ниже.
После сбора урожая в стадии зрелости растения, семена обезвоживают посредством хранения семян в течение 24 часов при комнатной температуре в эксикаторе, содержащем силикагель в качестве поглотителя влаги. Содержание влаги в семенах обычно составляет 6-8%. Общие липиды экстрагируют из известной массы обезвоженных семян посредством измельчения семян с помощью смеси хлороформа и метанола (2/1, об/об) в пробирке Эппендорфа, с использованием гомогенизатора для тканей Reicht (частота 22/секунду в течение 3 минут) и металлического шара. Добавляют один объем 0,1 М раствора KCl, и смесь встряхивают в течение 10 минут. Нижнюю неполярную фракцию собирают после центрифугирования смеси в течение 5 минут при скорости 3000 об/мин. Оставшуюся верхнюю (водную) фракцию промывают 2-мя объемами хлороформа при перемешивании в течение 10 минут. Вторую неполярную фракцию также собирают и объединяют в пул с ранее собранной. Растворитель испаряют из липидов в экстракте в потоке азота и высушенный общий липид растворяют в известном объеме хлороформа.
Для измерения количества липида в экстрагированном материале известное количество 17:0-TAG добавляют в качестве внутреннего стандарта и липиды из известного количества семян инкубируют в 1 н метанольной HCl (Supelco) в течение 2 часов при 80°C. Полученный таким образом FAME экстрагируют гексаном и анализируют способом GC. Количество отдельного FAME определяют на основании количества FAME 17:0 TAG. Массу отдельных FAME, после вычитания массы метильных групп, которыми этерифицированы FAME, переводят в моль посредством деления на молекулярную массу отдельных FAME. Общее количество моль всех FAME делят на три, чтобы вычислить количество моль TAG, и, таким образом, глицерина. Далее, количество моль TAG превращают в массу TAG. В конце, процент содержания масла на основе массы семени вычисляют с использованием массы семени, предполагая, что весь экстрагированный липид представляет собой TAG или эквивалент TAG для целей вычисления содержания масла. Данный способ базируется на Li et al., (2006). Другие семена подобного размера, кроме семян Camelina или рапса, также могут быть проанализированы этим способом.
Содержание масла в семени рапса и других семенах можно измерять способами ядерного магнитного резонанса (Rossell и Pritchard, 1991) пульсирующей волной NMS 100 Minispec (Bruker Pty Ltd Scientific Instruments, Germany). Способом ЯМР можно одновременно измерить содержание влаги. Кроме того, содержание масла в семенах можно измерить отражательной спектроскопией в ближнем ИК-диапазоне, например, с использованием монохроматора NIRSystems модель 5000. Кроме того, содержание влаги может быть измерено в образце партии семян, посредством сушки семян в образце в течение 18 часов при температуре приблизительно 100°C, согласно Li et al. (2006).
Анализ липидов из образцов лизата листа
Липиды из образцов лизата экстрагируют с использованием смеси хлороформ/метанол/0,1 М раствор KCl (2:1:1) и извлекают. Различные классы липидов в образцах разделяют на пластинках Силикагель 60 для тонкослойной хроматографии (TLC) (MERCK, Дармштадт, Германия) (TLC), пропитанных 10% раствором борной кислоты. Система растворителей, используемых для фракционирования TAG из экстракта липидов, представляет собой хлороформ/ацетон (90:10, об/об). Отдельные классы липидов визуализируют посредством контакта пластинок с парами йода и идентифицируют посредством параллельного анализа аутентичных стандартов на такой же пластинке TLC. Пластинки приводят в контакт с фосфорными визуализационными экранами на протяжении ночи и анализируют с помощью устройства для визуализации фосфора Fujifilm FLA-5000 перед жидкостно-сцинтиляционным подсчетом для количественного определения импульсов в минуту.
Выделение общего липида и фракционирование липидов из вегетативных тканей
Состав жирных кислот общего липида в образцах листьев и других вегетативных тканей определяли прямым метилированием жирных кислот в лиофилизированных образцах. Для количественной оценки общего липида, жирные кислоты в известной массе лиофилизированных образцов непосредственно метилируют с 17:0 СFA. Для определения уровней общих TAG в образцах листьев, TAG из экстрагированного общего липида фракционируют с помощью TLC и метилируют в присутствии внутреннего стандарта 17:0 TAG, вследствие присутствия в листьях значительных количеств полярных липидов. Это осуществляют, как указано ниже. Ткани, включая образцы листа, лиофилизируют, взвешивают (сухая масса) и общие липиды, экстрагированные как описано Bligh и Dyer (1959) или с использованием смеси хлороформ/метанол/0,1 М раствор KCl (ХМК; 2:1:1) в качестве растворителя. Общие липиды извлекают из образцов листа N. benthamiana после лиофилизации, добавляя 900 мкл смеси хлороформ/метанол (2:1, об/об) на образец листа диаметром 1 см. Добавляют 0,8 мкг DAGЭ на 0,5 мг массы сухого листа в качестве внутреннего стандарта, если должен быть проведен анализ TLC-ПИД. Образцы гомогенизируют с помощью гомогенизатора для тканей ULTRA-TURRAX производства IKA, после чего добавляют 500 мкл 0,1 М раствора KCl. Образцы обрабатывают вихревым перемешиванием, центрифугируют в течение 5 минут, и нижнюю фракцию собирают. Оставшуюся верхнюю фракцию экстрагируют повторно, добавляя 600 мкл хлороформа, обрабатывают вихревым перемешиванием и центрифугируют в течение 5 минут. Нижнюю фракцию извлекают и объединяют в пул с ранее собранной. Липиды сушат в потоке азота и ресуспендируют в 2 мкл хлороформа на мг сухой массы листа. Общие липиды листьев N. tabacum или образцов листа извлекают, как описано выше, с некоторыми модификациями. Если 4 или 6 листовых дисков (каждый с площадью поверхности приблизительно 1 см2) объединяют, то используют 1,6 мл растворителя ХМК, а если объединяют 3 или менее листовых дисков, то используют 1,2 мл ХМК. Лиофилизированные ткани листа гомогенизируют в пробирке Эппендорфа, содержащей металлический шар, с помощью гомогенизатора для тканей Reicht (Qiagen) в течение 3 минут при частоте 20/секунду.
Отделение нейтральных липидов способами TLC и переметилирования
Известные объемы полных экстрактов листа, например, 30 мкл, помещают на пластинку TLC силикагель 60 (1×20 см) (Merck KGaA, Германия). Нейтральные липиды фракционируют на различные типы и отделяют от полярных липидов способом TLC в уравновешенной емкости для проявления, содержащей систему растворителей гексан/диэтиловый эфир (DEE)/уксусная кислота (70:30:1, об/об/об). Полосы TAG визуализируют посредством обрызгивания примулином, маркируют в УФ-свете, соскабливают с пластины TLC, переносят во флаконы для GC емкостью 2 мл и сушат с помощью N2. Добавляют 750 мкл 1 н метанольного HCl (Supelco analytical, США) в каждый флакон, вместе с известным количеством C17:0 TAG в качестве внутреннего стандарта, в зависимости от количества TAG в каждом образце. Обычно, добавляют 30 мкг внутреннего стандарта для образцов с низким содержанием TAG, тогда как в случае образцов с высоким содержанием TAG применяют до 200 мкг внутреннего стандарта.
Образцы липида для анализа состава жирных кислот способом GC переметилируют посредством инкубирования смесей при 80°C в течение 2 часов в присутствии метанольного HCl. После охлаждения образцов до комнатной температуры, реакцию останавливают, добавляя 350 мкл H2O. Метиловые эфиры жирных кислот (FAME) извлекают из смеси, добавляя 350 мкл гексана, обрабатывая вихревым перемешиванием и центрифугируя со скоростью 1700 об/мин в течение 5 минут. Верхнюю гексановую фракцию собирают и переносят во флаконы для GC с коническими вставками емкостью 300 мкл. После выпаривания, образцы ресуспендируют в 30 мкл гексана. По 1 мкл вводят в прибор для GC.
Количество индивидуальных и общих жирных кислот (TFA) в липидных фракциях определяют с помощью GC, определяя площадь каждого пика, и вычисляя посредством сравнения с площадью пика для известного количества внутреннего стандарта. Содержание TAG в листе вычисляют как сумму глицерильных и жирных ацильных фрагментов во фракции TAG, с использованием соотношения: %, масс. TAG=100 x ((41 x общее количество моль FAME/3)+(общее количество FAME в г - (15 x общее количество моль FAME)))/грамм сухой массы листа, где 41 и 15 представляют собой молекулярную массу глицерильного фрагмента и метильной группы, соответственно.
Капиллярная газожидкостная хроматография (GC)
FAME анализируют способом GC с использованием GC Agilent Technologies 7890A (Пало-Альто, Калифорния, США), оборудованного колонкой SGE BPX70 (70% цианопропил полисилфенилен-силоксан) (30 м x 0,25 мм внутренний диаметр, толщина пленки 0,25 мкм), ПИД, инжектором с расщеплением/без расщепления и аутосемплером с инжектором Agilent Technologies 7693 Series. Гелий используют в качестве газа-носителя. Образцы вводят в режиме расщепления (соотношение 50:1) при температуре печи 150°C. После инжекции температуру печи удерживают на уровне 150°C в течение 1 минуты, затем повышают до 210°C со скоростью 3°C/минуту-1, и в конце до 240°C со скоростью 50°C/минуту-1. Пики количественно измеряют с помощью программного обеспечения Agilent Technologies ChemStation (Rev B.04.03 (16), Пало-Альто, Калифорния, США), на основании ответа известного количества внешнего стандарта GLC-411 (Nucheck) и внутреннего стандарта C17:0-Me.
Количественное определение TAG с помощью Iatroscan
1 мкл экстракта липида помещают на один Chromarod-SII для TLC-ПИД Iatroscan ™ (Mitsubishi Chemical Medience Corporation - Japan). Далее стойку Chromarod переносят в уравновешенную емкость для проявления, содержащую 70 мл системы растворителей гексан/CHCl3 /2-пропанол/муравьиная кислота (85: 10,716:0,567:0,0567, об/об/об/об). После инкубации в течение 30 минут, стойку Chromarod сушат в течение 3 минут при 100°C и немедленно сканируют на анализаторе TLC-ПИД Iatroscan MK-6s (Mitsubishi Chemical Medience Corporation - Japan). Площадь пиков внутреннего стандарта DAGЭ и TAG интегрируют с помощью программного обеспечения для интеграции SIC-480II (версия: 7.0-E, SIC System instruments Co., LTD - Japan).
Количественное определение TAG осуществляют в две стадии. Сначала во всех образцах сканируют DAGЭ для коррекции выходов экстракции, после чего концентрированные образцы TAG выбирают и разбавляют. Затем, количество TAG определяют в разбавленных образцах вторым сканированием в соответствии с внешней калибровкой, используя глицерилтрилинолеат в качестве внешнего стандарта (Sigma-Aldrich).
Площадь пика индивидуального FAME сначала корректируют на основании площади пика известных количеств таких же FAME, присутствующих в коммерческом стандарте GLC-411 (NU-CHEK PREP, Inc., USA). Скорректированные значения площади пика используют для вычисления массы каждого FAME в образце, по сравнению с внутренним стандартом. Поскольку масло хранится в основном в форме TAG, количество масла вычисляют на основании количества FAME в каждом образце. Общее количество моль глицерина определяют, вычисляя количество моль FAME, с последующим делением общего количества моль FAME на 3 Количество TAG вычисляют как сумму глицерильных и жирных ацильных фрагментов, с использованием формулы: %, масс. масла=100 x ((41 x общее количество моль FAME/3)+(общее количество г FAME - (15 x общее количество моль FAME)))/грамм сухой массы листа, где 41 и 15 представляют собой молекулярную массу глицерильного фрагмента и метильной группы, соответственно.
Экстракция и количественная оценка растворимого белка
Растворимый белок экстрагировали из 10-20 мг измельченной свежей ткани растений. Кратко, хлорофилл и растворимые сахара экстрагировали при 80°C в 50-80% (об./об.) этаноле в 2,5 мМ буфере HEPES при pH 7,5, и осадок сохраняли для определения количества растворимого белка. Осадок промывали в дистиллированной воде, ресуспендировали в 400 мкл 0,1 M NaOH и нагревали при 95°C в течение 30 мин. Количество растворимого белка в супернатанте определяли с использованием анализа Бредфорд (Bradford, 1976). Растворимый белок экстрагировали также из свежеизмельченной ткани в буфере, содержащем 100 мМ Tris-HCl pH 8,0 и 10 мМ MgCl2. При количественной оценке растворимого белка посредством анализа Бредфорд получили результаты, сходные с результатами, полученными с использованием экстракции с использованием NaOH.
Электрофорез в SDS-полиакриламидном геле (SDS-PAGE)
Общий белок экстрагировали из замороженной, измельченной ткани листа посредством нагревания образцов в буфере Лэмли (1:3 масс./об.) при 95°C в течение 10 мин. Аликвоты супернатанта, нормализованные по массе в свежем состоянии (FW), разделяли в 10% акриламидном геле в соответствии с Laemmli (1970).
Содержание азота в листьях
Общее содержание азота (% сухой массы, DW) в 2-2,2 мг лиофилизированной ткани листа определяли с использованием масс-спектрометра изотопных соотношений Europa 20-20 с системой подготовки ANCA, содержащей трубку для сжигания и восстановления, функционирующую при 1000°C и 600°C, соответственно.
Содержание углерода и энергии
Содержание углерода и энергии рассчитывали на основании количества TAG, крахмала и общих углеводов в тканях листа дикого типа и трансгенного листа (% сухой массы листа). Уровни крахмала (% сухой массы листа) сначала переводили в глюкозные эквиваленты посредством умножения на коэффициент 180/162, чтобы принимать во внимание потери воды из-за связей цепей. Растворимые сахара определяли как различи между общим уровнем углевода и уровнем крахмала. Содержание углерода и энергии по TAG и растворимым сахарам рассчитывали на основании плотности энергии, молекулярной массы и содержания углерода триолеина (35114 кДж/моль; 885,4 г/моль; 57 моль C/моль) и глюкозы (28,3 кДж/моль; 180 г/моль; 6 моль C/моль), соответственно. Содержание углерода и энергии по крахмал-глюкозным эквивалентам рассчитывали, как описано выше для фракции растворимых сахаров. В общем, формулы, использованные для получения содержания углерода и плотности энергии различных метаболических пулов углерода являются следующими:
Содержание углерода TAG (ммоль C/ г сухой массы листа)=(% TAG x 57 моль C/моль TAG x 1000)/(100×885,4 г/моль TAG)
Содержание углерода растворимых сахаров (ммоль C/ г сухой массы листа)=[(% общих углеводов - (% крахмала x 180/162)) x 6 моль C/моль глюкозы * 1000]/(100 * 180 г/моль глюкозы)
Содержание углерода крахмала (ммоль C/г сухой массы листа)=[(% крахмал x 180/162)) x 6 моль C/моль глюкозы * 1000]/(100 * 180 г/моль глюкозы)
Содержание энергии TAG (кДж/г сухой массы листа)=(% TAG x 39,66 кДж/г TAG)/100
Содержание энергии растворимых сахаров (кДж/г сухой массы листа)=[(% общих углеводов - (% крахмала x 180/162)) x 15,57 кДж/моль глюкозы]/100
Содержание энергии крахмала (кДж/г сухой массы листа)=[(% крахмала x 180/162)) x 15,57 кДж/моль глюкозы]/100.
Пример 2. Сайленсинг TAG-липазы в растениях, накапливающих высокие уровни TAG в ткани листа
Сахарозависимая TAG-липаза 1 (SDP1) продемонстрировала свою роль в обороте TAG в несеменных тканях A. thaliana, а также в ходе прорастания семени (Eastmond et al., 2006; Kelly et al., 2011; Kelly et al., 2013). SDP1 экспрессируется в развивающемся семени, а полипептид SDP1 дополнительно присутствует в зрелом семени в связи с масляными тельцами (но не в качестве покрытия). Сайленсинг гена, кодирующего SDP1, приводил к небольшому, но значимому увеличению уровней TAG в корнях и стеблях A. thaliana (< 0,4%, в пересчете на сухую массу), в то время как еще менее выраженное увеличение наблюдается в ткани листа (Kelly et al., 2013).
Для определения возможности дальнейшего повышения уровней TAG в тканях листа и стебля, по сравнению с соэкспрессией AtWRI1 и AtDGAT1, разработан эксперимент с сайленсингом эндогенного гена SDP1 в растениях N. tabacum, которые являются гомозиготными по T-ДНК, содержащей гены для трансгенной экспрессии полипептидов WRI, DGAT1 и олеозина (Vanhercke et al., 2014). Поиск BLAST по транскриптоме N. benthamiana (Naim et al., 2012) с использованием нуклеотидной последовательности AtSDP1 в качестве запроса идентифицировал транскрипт (Nbv5tr6385200, SEQ ID NO: 173) с гомологией в отношении гена SDP1 A. thaliana. Участок (SEQ ID NO: 174) размером 713 пар оснований выбран для опосредованного шпилькой сайленсинга гена. Сконструирован синтетический фрагмент размером 3,903 тыс. пар оснований на основе вектора pHELLSGATE12, который содержит, в приведенном порядке, конститутивный промотор enTCUP2, фрагмент SDP1 N. benthamiana размером 713 пар оснований в смысловой ориентации, фланкированный сайтами attB1 и attB2, интрон Pdk, последовательность интрона cat в обратной ориентации, второй фрагмент SDP1 N. benthamiana размером 713 пар оснований, фланкированный сайтами attB1 и attB2, в обратной (антисмысловой) ориентации, и участок терминатора/сайт полиаденилирования OCS 3' (Фигура 2). Вставку субклонируют в pJP3303 с использованием рестрикционных сайтов SmaI и KasI, где полученный вектор экспрессии обозначен pOIL051. Данная химерная ДНК содержит ген селективного маркера, обеспечивающий устойчивость к гигромицину.
pOIL051 применяют для получения трансформированных растений N. tabacum посредством опосредованной Agrobacterium трансформации. Исходные клетки растений получают от трансгенных растений, гомозиготных по T-ДНК pJP3502 (Vanhercke et al., 2014). Трансгенные растения, содержащие T-ДНК из pOIL051, отбирают по устойчивости к гигромицину и переносят в грунт в теплице или в комнату с контролируемой средой для продолжения роста. В ходе развития растения образцы листьев собирают с подтвержденных двойных трансформантов (Растения T0) до цветения, в период цветения и на стадии завязывания семян, и в каждом определяют уровень TAG. Трансгенные растения, содержащие только низкие уровни TAG в листьях или содержащие TAG на том же уровне, что и контрольные, идентифицируют с помощью экстракции липида из образцов листьев и анализа пятен TLC и отбрасывают. Уровни TAG в оставшейся популяции трансформантов количественно определяют способом GC, как описано в Примере 1. До цветения большинство из указанных растений демонстрируют значительно повышенные уровни TAG (> 5% от сухой массы листьев) в ткани листа, в то время как в 4 растениях присутствовали уровни TAG выше 10% (Таблица 3). Максимальный уровень TAG, наблюдаемый в листьях указанных растений, до цветения, составил 11,3% в растении 51-13. Для сравнения, трансгенные растения материнской линии N. tabacum, экспрессирующие AtWRI1, AtDGAT1 и олеозин, демонстрирует уровни TAG приблизительно 2% до цветения и приблизительно 6% в ходе цветения (Vanhercke et al., 2014). Таким образом, добавление SDP1-ингибирующей конструкции к комбинации AtWRI1 плюс AtDGAT1 является синергическим с точки зрения повышения уровней TAG в этих растениях. Неожиданно было обнаружено, что содержание TAG в листьях, собранных с двойных трансформированных растений в стадии цветения, было значительно повышено, составляя 30,5%, в пересчете на сухую массу (Таблица 4), что представляет собой увеличение в 5 раз относительно растений без сайленсинга SDP1. К большому удивлению авторов изобретения, уровень TAG достиг потрясающего значения 70,7% (% от сухой массы) в образцах стареющих листьев (зеленых и желтых) на стадии завязывания семян (Таблица 13). Применение ЯМР для измерения содержания масла в цельных листьях растений табака на стадии завязывания семян, содержание TAG в некоторых зеленых листьях, начавших стареть, составило приблизительно 43%, а в некоторых коричневых, дегидратированных листьях составило 42%. Если такие листья зажать между двумя коричневыми бумажными фильтрами, выделяющееся масло впитывается в бумагу и делает ее просвечивающейся, тогда как для листьев контрольного табака этого не наблюдалось, что предлагает простой способ скрининга для обнаружения растений с высоким содержанием масла.
Два первичных трансформанта (№61, №69), каждый из содержал Т-ДНК из pJP3502 и pOIL51 и демонстрировал высокие уровни TAG, проанализированы цифровой капельной ПЦР (цкПЦР) с использованием специфической для гена гигромицина пары праймеров, с целью определения количества инсерций T-ДНК pOIL51. Растение, обозначенное №61, содержит одну инсерцию T-ДНК из pOIL51, тогда как растение №69 содержит три инсерции T-ДНК из pOIL51. Проведен повторный скрининг растений-потомков T1 обеих линий способом цкПЦР с целью идентификации гомозиготных, гетерозиготных и нулевых растений. Растения-потомки растения №61, не содержащие инсерций из pOIL51 (нулевые; всего 7) или 2 инсерции T-ДНК (т.е., гомозиготные по указанной T-ДНК; всего 12) отобраны для дальнейшего анализа. Кроме того, растения-потомки линии №69, содержащие инсерции нулевые T-ДНК от pOIL51 (нуль; всего 2) или 2 такие инсерции (всего 15) или 4 или 5 инсерций (всего 5) сохраняли для дальнейшего анализа.
Отобранные растения T1 выращены в теплице в то же время и при тех же условиях, что и контрольные растения. Образцы ткани зеленых листьев растений T1, собранные до цветения, сушат и определяют содержание общих жирных кислот (TFA) и TAG способом GC. Содержание TFA в растениях, содержащих обе Т-ДНК, варьирует от 4,6% до 16,1%, в пересчете на сухую массу, в том числе уровни TAG в тех же листьях от 1,2% до 11,8%, в пересчете на сухую массу (Фигура 3). Это намного больше, чем в растениях, содержащих только T-ДНК из pJP3502, выращенных бок-о-бок в таких же условиях и проанализированных на такой же стадии роста, что снова демонстрирует синергию между снижением активности TAG-липазы и комбинации WRI1 плюс DGAT. В растениях, содержащих только T-ДНК pJP3502, присутствует от 4,2% до 6,8% TFA, в том числе уровни TAG от 1,4% до 4,1%, в пересчете на сухую массу (Фигура 3). Содержание в растениях дикого типа в среднем составляет приблизительно 0,8% TFA, в том числе менее чем 0,5% TAG, в пересчете на сухую массу. Состав жирных кислот в общем содержании жирных кислот и содержание TAG в листьях каждой из линий, №61 и №69, является сходным с их составом в листьях, содержащих только T-ДНК из pJP3502 (материнский). По сравнению с контрольными листьями дикого типа, растения, содержащие Т-ДНК из pOIL51 и pJP3502, демонстрируют повышенные уровни C16:0, C18:1 и C18:2 жирных кислот. Такой существенный сдвиг состава жирных кислот в значительной степени происходит за счет C18:3, содержание которой уменьшается с приблизительно 50-55% до приблизительно 20-30%, как процент от общего содержания жирных кислот.
Таблица 3. Уровни TAG (% сухой массы листа) и состав жирных кислот TAG в ткани листа растений N. tabacum (поколение T0), экспрессирующих трансгены WRI1, DGAT1 и олеозина и супертрансформированных T-ДНК, кодирующей конструкцию шпильки SDP1 (pOIL051), по сравнению с диким типом (нетрансформированным). Образцы листьев собраны в течение вегетативной стадии (до цветения). Образцы липида дополнительно содержали 0,0-0,2% C16:3, 0,0-0,4% C20:1; 0,0-0,1% C20:2n-6.
Таблица 4. Уровни TAG и состав TAG (% от сухой массы листа) в ткани листа растений N. tabacum (Поколение T0), экспрессирующих трансгены WRI1, DGAT1 и олеозина и супертрансформированных T-ДНК, кодирующей конструкцию шпильки SDP1 (pOIL051). Образцы листьев собраны в период цветения.
Таблица 5. Уровни TAG и состав TAG (% от сухой массы листа) в ткани листа растений N. tabacum (Поколение T0), экспрессирующих трансгены WRI1, DGAT1 и олеозина и супертрансформированных T-ДНК, кодирующей конструкцию шпильки SDP1 (pOIL051). Образцы листьев собраны в стадии завязывания семян. Y=желтый лист, G =зеленый лист.
Значительное увеличение уровней TFA, включая уровни TAG, между растениями, содержащими только T-ДНК pJP3502, и растениями, содержащими Т-ДНК из pOIL51 и pJP3502, сохраняется на протяжении развития растения. Контрольные растения, содержащие только T-ДНК из pJP3502, содержат от 7,7% до 17,5% TAG в ходе цветения, в то время как в период завязывания семян уровни TAG варьируют от 14,1% до 20,7%, в пересчете на сухую массу. Содержание TAG в листьях растений, содержащих Т-ДНК pJP3502 и pOIL51, варьирует от 6,3% до 23,3% в период цветения и от 12,6% до 33,6% в период завязывания семян. Подобные изменения состава жирных кислот во фракции TAG для обеих стадий были обнаружены, как было описано ранее для стадии вегетативного роста.
Кроме того, обнаружены повышенные уровни TAG и в других вегетативных тканях трансгенных растений, таких как корни и стебель. Некоторые ткани корня трансгенных растений N. tabacum, трансформированных T-ДНК pOIL051, содержат 4,4% TAG, а некоторые ткани стебля содержат 7,4% TAG, в пересчете на сухую массу (Фигура 10). Растения дикого типа и N. tabacum, содержащие только T-ДНК из pJP3502, демонстрируют намного более низкие уровни TAG в обоих видах тканей. Добавление конструкции шпильки SDP1 с целью снижения экспрессии эндогенной TAG-липазы оказывает очевидное синергическое влияние с генами, кодирующими фактор транскрипции и биосинтез TAG (WRI1 и DGAT), с точки зрения повышения содержания TAG в стеблях и корнях. Необходимо отметить, что более низкие уровни TAG в корнях сравнимы с тканями стебля в пределах одного и того же растения, в то время как в растениях дикого типа и N. tabacum, содержащие только T-ДНК из pJP3502 наблюдается обратная тенденция. Состав TAG в тканях корня и стебля демонстрирует изменения в содержании C18:1 и C18:3 жирных кислот, подобные наблюдаемым ранее в трансгенной ткани листа. Уровни C18:2 в TAG снижены в трансгенной ткани стебля, в то время как содержание C16 жирных кислот обычно снижено в трансгенных тканях корня, по сравнению с контролем дикого типа.
Таким образом, авторами изобретения сделан вывод о том, что добавление экзогенного гена к комбинации WRI1 и DGAT с целью сайленсинга эндогенного гена SDP1 повышает общее содержание жирных кислот, в том числе содержание TAG, на всех стадиях роста растения, и оказывает синергическое влияние с WRI1 и DGAT, особенно в стеблях и корнях. Семена T1 трансгенных растений помещают на питательные среды для тканей in vitro при комнатной температуре, чтобы проверить степень и расчет времени прорастания. Прорастание семени T1 трех независимо трансформированных линий по-прежнему сравнивают с семенем трансгенных растений, трансформированных T-ДНК из pJP3502. Кроме того, ранняя сила саженца, по-видимому, остается интактной. Это было неожиданным, с учетом роли SDP1 в прорастании семени A. thaliana и наблюдаемых дефектов при прорастании мутантов SDP1 (Eastmond et al., 2006). Для преодоления любых дефектов прорастания, если они возникнут, разработана вторая конструкция, в которой ингибирующая SDP1 РНК экспрессируется с промотора, который в семени по существу не экспрессируется или экспрессируется с низкими уровнями, например, такого как промотор из фотосинтетического гена, например, SSU. Авторы настоящего изобретения считают, что является предпочтительным, для уменьшения риска вредного влияния на прорастание семени или раннюю силу саженца, избегать конститутивного промотора для управления экспрессией РНК, ингибирующей SDP1, или по меньшей мере избегать промотора, экспрессирующегося в семенах.
Семена T1 трансгенных растений помещали на питательные среды для тканей in vitro при комнатной температуре, чтобы проверить степень и расчет времени прорастания. Прорастание семени T1 трех независимо трансформированных линий по-прежнему сравнивают с семенем трансгенных растений, трансформированных T-ДНК из pJP3502. Кроме того, ранняя сила саженца, по-видимому, остается интактной. Это было неожиданным, с учетом роли SDP1 в прорастании семени A. thaliana и наблюдаемых дефектов при прорастании мутантов SDP1 (Eastmond et al., 2006). Для преодоления любых дефектов прорастания, если они возникнут, разработана вторая конструкция, в которой ингибирующая SDP1 РНК экспрессируется с промотора, который в семени по существу не экспрессируется или экспрессируется с низкими уровнями, например, такого как промотор из фотосинтетического гена, например, SSU. Авторы настоящего изобретения что является предпочтительным, для уменьшения риска вредного влияния на прорастание семени или раннюю силу саженца, избегать конститутивного промотора, или по меньшей мере, избегать промотора, экспрессирующегося в семенах, для управления экспрессией РНК, ингибирующей SDP1.
Необходимо отметить, что растения T0 с наиболее высокими уровнями TAG выращены в условиях высокой освещенности в комнате с контролируемой средой (интенсивность света 500 микромоль, световой цикл 16 часов света/26°C-8 часов темноты/18°C), и они выглядели меньше (приблизительно 70% высоты относительно растений, трансформированных T-ДНК из pJP3502), чем контрольные растения дикого типа. Авторами изобретения сделан вывод о том, что комбинация трансгенов и/или генетических модификаций для подходов «push», «pull», «protect» и «package» особенно благоприятна для достижения высоких уровней TAG в вегетативных частях растения. В данном примере, WRI1 обеспечивает «push», DGAT обеспечивает «pull», сайленсинг SDP1 обеспечивает «protect», и олеозин обеспечивает «package» TAG.
Пример 3. Специфическая для старения экспрессия фактора транскрипции
Сообщалось, что эктопическая экспрессия основных регуляторов развития эмбриона и семени, например LEC2, повышает уровни TAG в несеменных тканях (Santos-Mendoza et al., 2005; Slocombe et al., 2009; Andrianov et al., 2010). Однако, повышенная экспрессия конститутивного LEC2 в растениях, трансформированных геном 35S-LEC2, приводит к нежелательному плеотропному влиянию на развитие и морфологию растения, включая соматический эмбриогенез и аномальные структура листа (Stone et al., 2001; Santos-Mendoza et al., 2005). Для оценки того, будет ограничение экспрессии LEC2 стадией старения листьев в ходе развития растения, т.е. после того, как растения полностью выросли и достигли полной биомассы, минимизировать нежелательные фенотипические эффекты, но по-прежнему увеличивать уровни липида в листьях, разработана и получена химерная ДНК для экспрессии LEC2 под контролем специфичного для старения промотора A. thaliana из гена SAG12 (U37336; Gan and Amasino, 1995).
Для получения генетической конструкции, получен синтетический фрагмент ДНК размером 3,635 тыс. пар оснований, содержащий, в указанном порядке, специфический для старения промотор SAG12 A. thaliana, последовательность, кодирующую белок LEC2 и участок терминатора/полиаденилирования гена лектина Glycine max. Указанный фрагмент вставляют между рестрикционными сайтами SacI и NotI pJP3303. Данная конструкция обозначена как pOIL049 и оценена в листьях растения N. tabacum, стабильно трансформированных генами, кодирующими полипептиды WRI1, DGAT1 и олеозина, содержащими T-ДНК из pJP3502. С применением опосредованных Agrobacterium способов трансформации, конструкцию pOIL049 используют для трансформации клеток растений N. tabacum, гомозиготных по T-ДНК pJP3502 (Пример 3). Трансгенные растения, содержащие гены из pOIL049, отбирают на основе устойчивости к гигромицину и выращивают в теплице до стадии зрелости. Образцы ткани трансгенных листьев получены на разных стадиях роста, включая старение листьев, и содержат повышенные уровни TAG, по сравнению с материнской линией N. tabacum pJP3502.
Всего получено 149 независимых растений T0 (т.е. первичных трансформантов). Cобраны образцы верхних зеленых листьев всех растений и нижних коричневых, полностью постаревших листьев выбранных событий на стадию завязывания семян в процессе развития растения, и содержание TAG количественно определено с помощью TLC-GC. Количество инсерций T-ДНК pOIL49 в отобранных растениях определено способом цкПЦР, с использованием пары праймеров, специфической в отношении гена гигромицина. Уровень TAG 30,2%, в пересчете на сухую массу, наблюдается в ткани зеленых листьев, собранной на стадии завязывания семян. Уровни TAG в коричневых листьях ниже в большинстве растений, от которых получены образцы. Однако, три растения (№32b, №8b и №23c) продемонстрировали более высокие уровни TAG в ткани постаревших коричневых листьев, чем в зеленых растущих листьях. Указанные растения содержали 1, 2 или 3 инсерции T-ДНК из pOIL49.
Проведен скрининг потомства T1 растений №23c и №32b способом цкПЦР с целью идентификации нулевых, гетерозиготных и гомозиготных по T-ДНК из pOIL049 растений. Растения-потомки растения №23c, содержащего нуль инсерций T-ДНК из pOIL049 (нулевые; всего 7) или две T-ДНК инсерции T-ДНК из pOIL049 (гомозиготные; всего 4) отобран для дальнейшего анализа. Кроме того, растения-потомки растения №32b, содержащие нуль инсерций (нулевые; всего 6) или две инсерции (гомозиготные; всего 9) сохранены для дальнейшего анализа. Образцы ткани зеленых листьев получены до цветения, и содержание TFA и TAG определено способом GC. Растения дикого типа и растения, трансформированные T-ДНК из pJP3502, были такими же как описано ранее (Пример 2), и были выращен бок-о-бок в одной теплице. Уровни TFA до цветения в листьях трансформантов, содержащих T-ДНК из pOIL049, варьируют от 5,2% до 19,5%, в пересчете на сухую массу (Фигура 5). Уровни TAG в этих же тканях варьируют от 0,8% до 15,4%, в пересчете на сухую массу. Это значительно больше, чем в растениях, содержащих только T-ДНК из pJP3502. Содержание TAG в растениях, содержащих Т-ДНК из pJP3502 и pOIL049, дополнительно повышается до уровня от 38,5% до 34,9% в период цветения и завязывания семян, соответственно. Когда состав жирных кислот в общем содержании жирных кислот был проанализирован для листьев, гомозиготных по T-ДНК из pOIL049, наблюдались повышенные уровни C18:2 и сниженные уровни C18:3 (Фигура 12), в то время как процентное содержание C16:0 и C18:1 остается приблизительно таким же, как и в листьях, трансформированных только T-ДНК из pJP3502. Указанные данные демонстрируют, что добавление второго гена фактора транскрипции под контролем неконститутивного промотора с целью обеспечения регулируемой развитием экспрессии может дополнительно повышать уровни TAG в вегетативных тканях растения. Кроме того, данные указывают на некоторую степень экспрессии специфичного для старения промотора SAG12 в зеленой ткани до старения листьев.
Уровни TAG значительно повышены в ткани стебля, по сравнению с растениями N. tabacum дикого типа и трансгенными растениями, содержащими только T-ДНК из pJP3502. Некоторые ткани стебля трансгенных растений N. tabacum, трансформированные T-ДНК из pOIL049, содержат 4,9% TAG, в пересчете на сухую массу (Фигура 6). С другой стороны, уровни TAG в ткани корня демонстрируют более выраженную вариацию между тремя растениями pOIL049, где некоторые ткани корня содержат 3,4% TAG. Необходимо отметить, что более низкие уровни TAG в корнях были сравнимыми с тканью стебля в пределах того же растения, в то время как обратная тенденция наблюдалась в растениях дикого типа и N. tabacum, содержащих только T-ДНК из pJP3502. Состав TAG в тканях корня и стебля демонстрирует изменения в содержании C18:1 и C18:3 жирных кислот, подобные наблюдаемым ранее в трансгенной ткани листа. Уровни C18:2 в TAG снижены в трансгенной ткани стебля, в то время как содержание C16 жирных кислот обычно снижено в трансгенных тканях корня, по сравнению с контролем дикого типа.
Получены соответствующие генетические конструкции, кодирующие другие факторы транскрипции под контролем промотора SAG12, а именно LEC1, LEC1like, FUS3, ABI3, ABI4, ABI5 и другие (Пример 9). Например, получены дополнительные конструкции для экспрессии фактора транскрипции однодольных LEC1 Zea mays (Shen et al., 2010) или LEC1 Sorghum bicolor (номер доступа Genbank XM_002452582.1) под контролем полученного из однодольных гомолога промотора SAG12 A. thaliana, такого как промотор SEE1 маиса (Robson et al., 2004). Дальнейшие конструкции получены для экспрессии факторов транскрипции под контролем регулируемых развитием промоторов, например, таких, которые предпочтительно, экспрессируются в период цветения (например, чувствительные к длительности светового дня промоторы), фитохромных промоторов, криптохромных промоторов, или в стеблях растения в ходе вторичного роста, таких как промотор из гена CesA. Указанные конструкции применяются для трансформации растений, где отбираются растения, которые продуцируют по меньшей мере 8% TAG в вегетативных частях.
Пример 4.Анализ трансгенных растений
Растительный материал и условия роста
Растения из трех накапливающих TAG трансгенных линий выращивали в ростовых камерах или в теплице в контролируемых условиях:
1. Растения с повышенной экспрессией генов, кодирующих WRI1, DGAT и олеозин (Vanhercke et al, 2014), обозначенные в настоящем описании как растения HO, представляли собой растения поколения T2, которые были гомозиготными по введенной T-ДНК из pJP3052.
2. Растения T1, трансформированне конструкцией РНКи для осуществления сайленсинга TAG-липаза SDP1, так же как T-ДНК из pJP3502, кодирующей полипептиды WRI1, DGAT и олеозин, из двух независимых трансформированных линий. См. Пример 2. Эти растения обозначены SDP1.
3. Растения T1, трансформированные генетической конструкцией для повышенной экспрессии фактора LEC2 с промотора SAG12, так же как T-ДНК из pJP3502, кодирующей полипептиды WRI1, DGAT и олеозин. См. Пример 3. Эти растения обозначены LEC2, и являлись потомством одного растения T0.
4. Растения дикого типа (WT, или культивар Wisconsin 38) использовали в кчестве контрольных растений и выращивали в то же время и в таких же условиях, как трансгенные растения. Для вегетативных образцов, растения табака WT и HO выращивали в ростовых камерах для растений PGC20/PGC20FLEX (Conviron) при концентрациях CO2 окружающей среды с освещением 250-450 мкмоль м-2 с-1 от флуоресцентных ламп. Растения выраивали с суточными циклами 12 час свет/25°C: 12 час темнота/20°C. Растения, предназначенные для отбора образцов на 49 сутки после посева (DAS), выращивали в 1,25-литровых горшках в грунте с удобрением осмокот. Растения, предназначенные для отбора образцов на 69 DAS, выращивали в 4-литровых горшках в грунте и поливали каждые 14 суток с удобрением аквазоль. Для всех анализов, образцы отбирали из четырех растений каждого генотипа. Для образцов, подлежащих сбору на стадии роста образованием семян, растения WT, HO, SDP1 и LEC2 выращивали в теплице без искусственного света (n=3, 3, 8, 6, соответственно). Для всех анализов, листовые диски собирали из листьев в конце фазы роста при свете, быстро замораживали и хранили при -80°C до анализа.
Уровни TAG и состав жирных кислот
Уровни TAG измеряли в листьях зрелых растений WT, HO, SDP1 и LEC2. Определяди также состав жирных кислот в TAG в листьях. Данные показаны в Таблице 6 для растений LEC2 и SDP1.
Уровни крахмала и сахаров
Уровни крахмала и растворимых сахаров измеряли в образцах ткани листа из растений дикого типа (WT) и трансгенных растений HO, SDP1 и LEC2. Как правило, в листьях, содержащих обе Т-ДНК, обнаруживали обратную корреляцию между уровнями TAG и крахмала в ткани листа, в пересчете на сухую массу (Фигура 7 и Фигура 8). И наоборот, в трансгенных растениях уровни растворимых сахаров в листьях являлись приблизительно таким же, как и в растениях дикого типа, что позволяет предполагать отсутствие значимого узкого места в переводе сахаров в TAG. Эффект положения листьев в растении наблюдали в растениях дикого типа, в которых уровни крахмала имели тенденцию к увеличению от более низкого к более высокому положению листьев. В трансгенных растениях не детектировали такого эффекта.
Содержание углерода и энергии
Содержание углерода и энергии в TAG, содержание крахмала и сахара в листьях растений HO, SDP1 и LEC2 измеряли и сравнивали с растениями дикого типа, в пересчете на сухую массу. Данные (Таблица 7) показали, что каждое из содержания углерода и энергиии увеличивалось в растениях HO и увеличивалось даже больше в растениях SDP1 и LEC2 ао сравнению с растениями WT. Увеличение наблюдали для суммарного содержания TAG, крахмала и растворимых сахаров, так же как для суммарного содержания TAG и крахмала. Заключили, что увеличение содержания углерода посредством увеличения содержания TAG более чем компенсировало уменьшенное содержание крахмала. Таким образом, для трансгенных растений показано увеличенное общее содержание углерода и увеличенное общее содержание энергии в пересчете на сухую массу.
Содержание азота и растворимого белка
Содержание азота и белка измеряли в образцах листьев трансформированных и контрольных растений, как описано в Примере 1, для растений на 69 DAS. Третий лист от верхушки каждого из растений табака WT и HO, листья которых еще не полностью развернулись и таким образом, еще продолжали расти, имели одинаковое содержание азота приблизительно 3,0% по DW. Более старые (нижние) листья на каждом растении также анализировали. В растениях WT, содержание азота в листьях уменьшалось с возрастом листа, в то время как содержание азота относительно сохранялось в более старых листьях HO с меньшим снижением, по сравнению с растениями WT plants. Например, более старые листья, такие как лист 11 от верхушки растений HO, содержали более чем в два раза больше азота (2,9%), по сравнению с соответствующими листьями в растениях WT plants (1,3%; Фигура 9a). Сходную тенденцию наблюдали для общего растворимого белка, где в два раза больше растворимого белка детектировали в более старых листья HO по сравнению с WT (10,4 и 5,0 мкг/мг FW, соответственно; Фигура 9B). Такие же тенденции наблюдали, когда образцы растворимого белка подвергали электрофорезу в SDS-PAGE, после нормализации загрузки образцов в соответствии с массой свежего листа.
Таблица 6. Уровни TAG (% сухой массы листа) и состав жирных кислот в TAG избранных первичных трансформантов N. tabacum со сверхэкспрессией LEC2 (pOIL049) или осуществляющей сайленсинг конструкции, нацеленной против гена, кодирующего TAG-липазу SDP1 (pOIL051). Обеими конструкциями независимо трансформировали полученную ранее трансгенную линию N. tabacum со сверхэкспрессией генов, кодирующих WRI1, DGAT1 и ОЛЕОЗИН (Vanhercke et al., 2014).
Таблица 7. Содержание углерода и энергии TAG, крахмала и растворимых сахаров в тканях листьев относящихся к дикому типу и трансгенных растений N. tabacum.
*: принимая 2803 кДж/моль для глюкозы и 35114 кДж/моль для триолеина (Sanjaya et al., 2011)
В растениях как WT, так и HO, содержание белка в листьях увеличивалось с возрастом растения (Фигура 10, Таблица 8). В боле молодых растениях (49 DAS), содержание растворимого белка было немного, но не значимо больше в более старых листьях из растений HO по сравнению с WT. На 69 DAS, это различие было значимым, с 87% увеличением в старых листьях HO, по сравнению с WT (p<0,05, t-критерий). Заключили, что листья растений HO имели значимо увеличенное содержание азота и белка, по сравнению с соответствующими листьями в растениях WT. В этом контексте, «соответствующий лист» означает лист такого же возраста из растения, выращенного в таких же условиях.
Таблица 8. Содержание растворимого белка в листьях табака WT и HO (мкг/мг FW). Диапазон включает молодые, зрелые и более старые листья от более молодых (49 DAS) и более старых (69 DAS) растений.
Содержание азота в растениях SDP1 и LEC2
Для трансгенных растениц, обозначенных LEC2 и SDP1, показано увеличенное накопление TAG, по сравнению с растениями HO в ходе роста (Примеры 2 и 3), увеличивающееся с возрастом растения. Образцы листьев трансгенных растений, выращенных в ростовых камерах или в теплице, анализировали по содержанию азота, белка и углерода. Для растений LEC2 и SDP1 показано каждое из увеличения содержания углерода в листьях, содержания азота в листьях и содержание растворимого белка в листьях, по сравнению с растениями WT (Таблица 9). На стадии роста с завязыванием семян, листья LEC2 и SDP1 имели между 50% и 100% больше азота, чем листья WT. Содержание растворимого белка в листьях увеличивалось между 40% и 87% в листьях LEC2 и SDP1, соответственно, по сравнению с листьями WT. Содержание углерода в листьях также было увеличено. Несмотря на умеренное увеличение содержания углерода в листьях (от 16% до 21%) в линиях LEC2 и SDP1, большее относительное увелмчение содержания азота в листьях уменьшало соотношение углерода к азоту на вплоть до 40%, по сравнению с листьями WT.
Анализ общего содержания пищевых волокон (TDF)
Анализ общего содержания пищевых волокон для WT, SDP1 и LEC2 в зрелых листьях, полученных при цветении, показал, что листья WT имели содержание TDF 27%, листья SDP1 имели содержание TDF 15,9% (уменьшение на 59%, по сравнению с WT), и листья LEC2 имели содержание TDF 17,9% (уменьшение на 34% по сравнению с WT).
Таблица 9. Содержание TAG (% сухой массы), содержание углерода, содержание азота и содержание растворимого белка в листьях табака WT, LEC2 и SDP1. Для анализа TAG n=3-8, и для C, N и растворимого белка n=2-5.
Повышающая регуляция генов, вовлеченных в фотосинтез
Описанные выше наблюдения увеличения содержания углерода и энергиии в трансгенных растениях привели авторов настоящего изобретения к рассмотрению того, могут ли растения иметь увеличенную способность к фотосинтезу, связанную с измененным распределением углерода между крахмалом и TAG. Таким образом, транскриптом растений HO определяли и сравнивали с транскриптомом растений WT, выращенных в таких же условиях. РНК выделяли из растений на стадии цветения, переводили в кДНК и определяли полные транскриптомы. Когда в полученных библиотеках последовательностей сравнивали частоту представленности индивидуальных генов, наблюдали повышающую регуляцию (повышенную экспрессию) многочисленных генов, вовлеченных в фотосинтез, в растениях HO. В Таблице 10 перечислены репрезентативные гены, подвергнутые повышающей регуляции. Из этого заключили, что способность к фотосинтезу увеичена в трансгенных растениях.
Эффекты модификации фотопериода и интенсивности освещения
Условия роста модифицировали по сравнению с описанными выше, чтобы протестировать эффект увеличения или уменьшения фотопериода от 12 час, и увеличения интенсивности освещения. В одной ростовой камере с использованием высокой интенсивности освещения и длинного фотопериода, концентрация CO2 также была увеличена по сравнению с окружающей средой. Тестировали следующие условия, в каждом случае, растения выращивали в ростовых камерах для растений PGC20/PGC20FLEX (Conviron) при 25°C во время периода света, при 20°C во время периода темноты, и образцы листьев собирали на стадии роста с завязыввнием семян из листьев номер 9, 15 и 20, считая снизу каждого растения. Лист 9, таким образом, являлся самым старым из образцов листьев, лист 15 - промежуточным, и лист 20 - самым молодым из образцов листьев. В листьях анализировали общее содержание жирных кислот (TFA), как описано в Примере 1.
Таблица 10. Список генов, которые связаны с фотосинтезом и экспрессия которых подвергается повышающей регуляции.
_i2
_i2
_i6
_i1
_i2_1
_i1_1
_i1
_i1
_i3
_i1
_i1
_i1_1
_i3
_i1
_i2_1
_i1
_i1
_i1_1
_i1
_i1
_i5
_i1
_i3_1
_i1_2
_i2
_i1
_i1_1
1. Контрольные условия: 8 растений выращивали при освещенности 300 мкмоль м-2 с-1 от флуоресцентных ламп, с 12-часовым фотопериодом;
2. Увеличенная интенсивность освещения: 7 растений выращивали при освещенности 700 мкмоль м-2 с-1 от флуоресцентных ламп, с 12-часовым фотопериодом;
3. Уменьшенный фотопериод: 9 растений выращивали при освещенности 700 мкмоль м-2 с-1 от флуоресцентных ламп, с 8-часовым фотопериодом;
4. Увеличенные интенсивность освещения и фотопериод: 10 растений выращивали при освещенности 700 мкмоль м-2 с-1 от флуоресцентных ламп, с 12-часовым фотопериодом, при концентрации CO2 700 м.д.
Средние данные для листьев 9, 15 и 20 для каждого генотипа нанесены на график на Фигуре 11. Только увеличенная интенсивность освещения значительно не влияла на уровни TFA. Уменьшение фотопериода с 12 час до 8 час уменьшало уровни TFA, но в неожиданно малой степени. То есть, даже уменьшение количества света, получаемого каждые 24 часа, на 33%, оказывало заметно небольшой эффект. Наиболее заметыми из наблюдаемых результатов являлись результаты теста с использованием увеличенного фотопериода при уменьшенной интенсивности освещения и увеличенной концентрации CO2. Уровни TFA были значительно увеличены, достигая 50% (масс./масс. сухой массы) и выше в листьях растений LEC2. Поскоьку с использованием анализов TFA измеряли только компоненты жирных кислот из липидов, это означает, что общий уровень липидов был даже выше в этих листьях.
Пример 5. Модифицированные признаки в вегетативных частях однодольных растений
Химерные конструкции ДНК разработаны таким образом, чтобы повысить содержание масла в однодольных растениях, например, растении C4 S. bicolor (сорго), посредством экспрессии комбинации генов, кодирующих WRI1, LEC1 Z. mays (номер доступа AAK95562; SEQ ID NO: 155), DGAT и олеозина в трансгенных растениях. Несколько пар конструкций для биолистической сотрансформации разработаны и получены посредством лигирования-клонирования рестрикционного фермента, как указано ниже.
Генетическая конструкция pOIL136 представляет собой бинарный вектор, содержащий три кассеты экспрессии однодольных, а именно ген селективного маркера, кодирующий фосфинотрицин ацетилтрансферазу (PAT) для отбора растения, вторую кассету для экспрессии DGAT и третью для экспрессии олеозина. Сначала получают pJP136 амплификацией промотора гена актина из Oryza sativa (McElroy et al., 1990) и инсерции его в качестве фрагмента тупого-ClaI в pORE04 (Coutu et al., 2007) с получением pOIL094. Далее получают pOIL095 посредством инсерции версии гена олеозина Sesamum indicum, кодон-оптимизированного для экспрессии в однодольных, в pOIL094 на сайте KpnI. pOIL093 получают клонированием версии кодон-оптимизированного для экспрессии в однодольных гена DGAT2a Umbelopsis ramanniana (Lardizabal et al., 2008) в качестве фрагмента SmaI-KpnI в вектор, уже содержащий промотор гена убиквитина Zea mays. Далее получают pOIL134 клонированием кассеты экспрессии NotI DGAT2a из pOIL093 в pOIL095 на сайтах NotI. pOIL141 получают инсерцией гена селективного маркера, кодирующего PAT в качестве фрагмента BamHI-SacI в вектор, содержащий промотор убиквитина Z. mays. В конце получают pOIL136 клонированием кассеты экспрессии убиквитина Z. mays::PAT в качестве фрагмента тупого-AscI в ZraI-AscI pOIL096. Генетическая конструкция pOIL136, таким образом, содержит следующие кассеты экспрессии: промотор актина O. sativa::олеозин S. indicum, промотор убиквитина Z. mays::DGAT2a U. ramanniana и промотор убиквитина Z. mays::PAT.
Подобный вектор pOIL197, содержащий NPTII вместо PAT, конструируют субклонированием кассеты убиквитина Z. mays::NPTII из pUKN в качестве фрагмента HindIII-SmaI в сайты AscI (тупые) и HindIII pJP3343. Далее полученный вектор, pOIL196, расщепляют с помощью HindIII (тупые) и AgeI. Полученный фрагмент размером 3358 пар оснований клонируют в сайты ZraI-AgeI pOIL134 с получением pOIL197.
Разработан и получен набор конструкций, содержащих гены, которые кодируют WRI1 Z. mays (ZmWRI) или факторы транскрипции LEC1 (ZmLEC1) под контролем различных промоторов, для биолистической сотрансформации в комбинации с pOIL136 или pOIL197, с целью оценки влияния активности промотора и специфичности в отношении клетки на функционирование WRI1 или LEC1, или обоих в случае комбинации, при экспрессии в вегетативных тканях растения C4, такого как сорго. Указанный отдельный набор конструкций не содержит гена селективного маркера, за исключением pOIL333, который содержит NPTII в качестве селективного маркера. Различные исследуемые промоторы были такими, как указано ниже. Промотор гена убиквитина Z. Mays (pZmUbi) представляет собой активный конститутивный промотор однодольных, в то время как о расширенном промотор CaMV 35S (e35S), содержащем двойной участок энхансера, сообщалось, что он приводит к более низким уровням экспрессии трансгена (обзор в Girijashankar и Swathisree, 2009). Тогда как промотор гена фосфоенолпируваткарбоксилазы (pZmPEPC) Z. mays активен в мезофильных клетках листа (Matsuoka и Minami, 1989), месте фотосинтеза в видах растений C4, промотор гена маленькой субъединицы Z. mays Rubisco (pZmSSU) является специфическим для слоя клеток обкладки сосудистых пучков (Nomura et al., 2000; Lebrun et al., 1987), т.е. клеток, в которых имеет место фиксация углерода в растениях C4.
Экспрессия гена Z. mays, кодирующего цистеиновую протеазу SEE1 (номер доступа AJ494982) идентифицирована, как сходная с экспрессией специфичного для старения промотора SAG12 A. thaliana в ходе развития растения. Таким образом, промотор размером 1970 пар оснований из гена SEE1 (SEQ ID NO: 207) был дополнительно выбран для управления экспрессией генов, кодирующих факторы транскрипции WRI1 и LEC1 Z. mays. Далее, промотор гена, кодирующего белок «цинкового пальца» AlSAP Aeluropus littoralis (Ben Saad et al., 2011; номер доступа DQ885219; SEQ ID NO: 208), промотор гена, кодирующего DIRIGENT (DIR16) гибрида Saccharum (Damaj et al., 2010; номер доступа GU062718; SEQ ID NO:246), промотор гена, кодирующего O-метил-трансферазу гибрида Saccharum (OMT) (Damaj et al., 2010; номер доступа GU062719; SEQ ID NO:247), аллель A1 промотора гена, кодирующего R1MYB1 гибрида Saccharum (Mudge et al., 2009; номер доступа JX514703,1; SEQ ID NO:248), промотор гена, кодирующего ген нагрузки стебля 5 (LSG5) гибрида Saccharum (Moyle and Birch, 2013; номер доступа JX514698.1; SEQ ID NO:249) и промотор чувствительного к сахарозе гена ArRolC A. rhizogenes (Yokoyama et al., 1994; номер доступа DQ160187; SEQ ID NO:209) дополнительно выбраны для экспрессии ZmWRI1 в ткани стебля. Таким образом, каждый из этих промоторов индивидуально присоединяли выше кодирующих областей ZmWRI1 или ZmLEC1, как указано ниже.
Промежуточный вектор, pOIL100, сначала получают клонированием последовательности, кодирующей WRI1 Z. mays, и участка терминатора транскрипции/полиаденилирования, фланкированных сайтами AscI-NcоI, в такие же сайты в бинарном векторе pJP3343. Различные версии конструкций для экспрессии WRI1 основаны на данном векторе и получены клонированием различных промоторов в pOIL100. pOIL101 получают клонированием фрагмента XhoI-Sal, содержащего промотор e35S с удвоенным участком энхансера, в сайт XhoI pOIL100. pOIL102 получают клонированием фрагмента HindIII-AvrII, содержащего промотор гена убиквитина Z. mays, в сайты HindIII-XbaI pOIL100. pOIL103 получают клонированием фрагмента HindIII-NcoI, содержащего промотор гена PEPC Z. mays, в сайты HindIII-NcoI pOIL100. pOIL104 получают клонированием фрагмента HindIII-AvrII, содержащего промотор гена SSU Z. mays, в сайты HindIII-AvrII pOIL100.
Синтезирован синтетический фрагмент, содержащий промоторная область SEE1 Z. mays, фланкированный уникальными сайтами HindIII-XhoI. Данный фрагмент клонировали в направлении против хода транскрипции по отношению к участку, кодирующему белок WRI1 Z. mays, используя сайты HindIII-XhoI в pOIL100. Результирующий вектор обозначен pOIL329. Синтезирован содержащий синтетический фрагмент промоторная область AlSAP A. littoralis, фланкированный уникальными сайтами XhoI-XbaI. Данный фрагмент клонировали в направлении против хода транскрипции по отношению к участку, кодирующему WRI1 Z. mays, используя сайты XbaI-Xho I в pOIL100. Результирующий вектор обозначен pOIL330. Синтезирован содержащий синтетический фрагмент промоторная область ArRolC A. rhizogenes, фланкированный уникальными сайтами PspOMI-XhoI. Данный фрагмент клонируют в направлении против хода транскрипции по отношению к участку, кодирующему WRI1 Z. mays, используя сайты PspOMI-XhoI в pOIL100. Результирующий вектор обозначен pOIL335. В конце, бинарный вектор (pOIL333), содержащий кассету экспрессии SEE1 Z. mays::ZmLEC1, получают в три стадии. Сначала, вектор экспрессии 35S::GUS конструировали амплификацией участка, кодирующего GUS, с фланкирующими праймерами, содержащими сайты AvrII и KpnI. Далее полученный фрагмент клонируовали в сайты SpeI-KpnI pJP3343. Результирующий вектор обозначен pTV111. Затем, участок 35S промотора pTV111 заменяли промотором SEE1Z. mays. На этой стадии последовательность SEE1 Z. mays амплифицировали с использованием фланкирующих праймеров, содержащих уникальные сайты HindIII и XhoI. Результирующий фрагмент разрезали соответствующими рестрикционными ферментами и субклонировали в сайты SalI-HindIII pTV111. Результирующий вектор обозначен pOIL332. Далее последовательность, кодирующую ZmLEC1 амплифицировали с использованием фланкирующих праймеров, содержащих сайты NotI и EcoRV. Полученный фрагмент субклонировали в соответствующие сайты pOIL332 с получением pOIL333.
Синтезировали синтетический фрагмент 2673 пар оснований, содержащий промоторную область DIR16 Saccharum, фланкированную участками HindIII-XbaI. Этот фрагмент клонировали выше кодирующей области белка WRI1 Z. mays с использованием участков HindIII-XbaI в pOIL100. Полученный вектор обозначен pOIL337. Синтезировали синтетический фрагмент 2947 пар оснований, содержащий промоторную область Saccharum, фланкированную участками XhoI-XbaI. Этот фрагмент клонировали выше кодирующей области белка WRI1 Z. mays с использованием участков XhoI-XbaI в pOIL100. Полученный вектор обозначен pOIL339. Синтезировали синтетический фрагмент 1181 пар оснований, содержащий промоторную область R1MYB1 Saccharum, фланкированную участками HindIII-XhoI. Этот фрагмент клонировали выше кодирующей области белка WRI1 Z. mays с использованием участков HindIII-XhoI в pOIL100. Полученный вектор обозначен pOIL341. Синтезировали синтетический фрагмент 4482 пар оснований, содержащий промоторную область LSG5 Saccharum, фланкированную участками XbaIII-SmaI. Этот фрагмент клонировали как фрагмент XbaIII-SmaI выше кодирующей области белка WRI1 Z. mays с использованием участков StuI-NheI в pOIL100. Полученный вектор обозначен pOIL343.
Получали полноразмерную плазмидную ДНК pOIL101, pOIL102, pOIL103, pOIL104, pOIL197 и pOIL136 для биолистической трансформации. Затем ДНК pOIL197 смешивали с pOIL101, pOIL102, pOIL103 или pOIL104 и трансформировали посредством биолистической трансформации в дифференцирующиеся ткани эмбриональных каллюсов (DEC) S. bicolor (семя сорго TX430), как описано в Примере 1. Альтернативно, с использованием конструкций для экспрессии таких же комбинаций генов проводили отдельную трансформацию или совместную трансформацию тканей DEC с помощью опосредованной Agrobacterium трансформации (Gurel et al., 2009; Wu et al., 2014).
От двадцати пяти до пятидесяти трансгенных растений подвергали регенерации и отбору по устойчивости к антибиотику для пар конструкций, включающих pOIL197 с каждой из pOIL102 (pZmUbi::WRI1), pOIL103 (pZmPEPC::WRI1) и pOIL104 (pSSU::WRI1). Проводили также трансформацию с использованием pOIL197 отдельно и с использовать pOIL102 или pOIL103 отдельно, и для контроля с «пустым вектором». Присутствие желательных трансгенов в растениях, которые были устойчивыми к селективному средству, демонстрировали посредством ПЦР. Количество копий каждого трансгена также определяли посредством цифровой ПЦР.
Общее содержание липидов в листьях оценивали количественно в первой подгруппе трансгенных растений S. bicolor перед их переносом из среды MS в грунт. Этот предварительный скрининг позволял предполагать немного увеличенные общие уровни липидов в ткани листьев для некоторых событий трансформации нв этой очень ранней стадии. Линию с наивысшим общим содержанием липидов, pOIL136 (2), далее анализировали посредством тонкослойной хроматографии (TLC) для определения эффекта экспрессии трансгена на накопление TAG. Образцы ткани листа этой конкретной линии отбирали на вегетативной стадии после переноса в грунт в теплице. По сравнению с отрицательным контролем дикого типа и отрицательным контролем с пустым вектором, для pOIL136 (2) показаны увеличенные уровни TAG в ткани листа после разделения TLC и окрашивания иодом. Последующая количественная оценка выявила 10-кратное увеличение TAG в трансгенной линии по сравнению с отрицательным контролем. В профиле TAG преобладали полиненасыщенные жирные кислоты линолевая и α-линоленовая кислота.
После переноса подтвержденных трансгенных растений в грунт в горшках в теплице, отбирали образцы целых листьев первичных трансформантов на вегетативной стадии роста (т.е. до появления трубчатого листа), на стадии трубчатого листа (определенной как время, когда трубчатый лист появился полностью, трубчатый лист представляет собой последний лист, образующийся у растения, из которого появляется метелка (верхушка)) и на стадии зрелости с завязыванием семян. Содержание общих жирных кислот (TFA) и триацилглицерина (TAG) (% сухой массы листа) количественно оценивали посредством TLC-GC, как описано в Примере 1.
Уровни TFA для отрицательного контроля дикого типа и отрицательного контроля с пустым вектором уменьшались в ходе развития растения (Таблица 11) и лежали в диапазоне 0,7-3,3% (масс./сухая масса). Наивысшие уровни TFA детектированы перед появлением трубчатого листа (что называют вегетативной стадией роста) и не превышали 3,3%. Уровни TAG в тех же растениях были постоянно низкими, в диапазоне 0-0,2% в ходе всего жизненного цикла растения (Таблица 11). Как содержание TFA, так и содержание TAG имело состав жирных кислот с преобладанием C16:0, C18:29,12 (LA) и C18:39,12,15(ALA). В частности, ALA присутствовала в приблизительно 50-75% из содержания TFA, что отражает использование этой жирной кислоты в мембранах пластид дикого типа. ALA также являлась главной жирной кислотой в очень небольшом количестве TAG, присутствующем в листьях дикого типа.
Тридцать пять подтвержденных трансгенных растений, трансформированных с использованием pOIL197 или pOIL136, где каждый из векторов содержал оба гена pZmUbi:DGAT и pZmUbi:олеозин в дополнение к генам селективного маркера, анализировали на вегетативной стадии, стадии трубчатого листа и на стадии зрелости с завязыванием семян. Данные представлены в Таблицах 12-14. В общем, для первичных трансформантов pOIL197 и pOIL136 показано увеличенное накопление TFA и TAG, по сравнению с линиями отрицательного контроля, но только до приблизительно удвоения уровня TFA по сравнению с контролем. Наивысшие уровни TFA детектированы на вегетативной стадии роста (Таблица 12). Подобно линиям дикого типа и отрицательного контроля, уровни TFA уменьшались с увеличением возраста растения (Таблицы 13 и 14). Максимальные уровни TFA на вегетативной стадии, стадии трубчатого листа и на стадии зрелости с завязыванием семян равны 5%, 4,5% и 2,1%, соответственно. Наивысшие детектированные уровни TAG менялись между 0,9 и 1,9%, в зависимости от возраста растения на время сбора образцов (Таблица 13), таким образом, были увеличены вплоть до 10 раз, по сравнению с очень низкими уровнями в листьях дикого типа (Таблица 11). Состав TFA оставался по большей части неизменным на различных стадиях, и ALA преобладала. Для состава TAG показана более высокая степень изменчивости между различными трансгенными линиями. По сравнению с составом жирных кислот в содержании TFA, уровни стеариновой кислоты, олеиновой кислоты и LA (18:29,12) постоянно увеличивались в TAG на протяжении всех исследованных стадий развития растений.
Девять первичных трансгенных растений, полученных посредством трансформации с использованием pOIL102 (pZmUbi:WRI1), получали посредством совместной бомбардировки pOIL102 и pUKN, содержащей ген селективного маркера NPTII. В Таблицах 15-17 показаны данные для содержания TFA и TAG, и состава жирных кислот, измеренные на всех трех стадиях роста. По сравнению с растениями, трансформированными конструкциями, кодирующими DGAT2 и олеозин (pOIL197 или pOIL136), уровни TFA и TAG в трансгенных событиях для pOIL102 были в целом более низкими. Действительно, уровни TFA и TAG были сходными с уровнями в растениях дикого типа и отрицательного контроля. Максимальные уровни TFA на вегетативной стадии, стадии трубчатого листа и на стадии зрелости с завязыванием семян составляли 2,6%, 2,5% и 2,0%, соответственно (Таблицы 15-17). Максимальные наблюдаемые уровни TAG составляли 0,2%, 0,4% и 0,9% на вегетативной стадии, стадии трубчатого листа и на стадии зрелости с завязыванием семян, соответственно.
Тридцать шесть первичных трансгенных растений, полученных посредством совместной бомбардировки pOIL197 (pZmUbi:DGAT и pZmUbi:олеозин) и pOIL102 (pZmUbi:WRI1) и с подтвержденным наличием интегрированных обеих генетических конструкций, анализировали по содержанию TFA и TAG, и составу жирных кислот на всех трех стадиях роста. Данные представлены в Таблицах 18-20. Некоторые растения имели сильно увеличенные уровни TFA и TAG по сравнению с трансформацией отдельными векторами pOIL197, pOIL136 или pOIL102. Максимальные уровни TFA на вегетативной стадии, стадии трубчатого листа и на стадии зрелости с завязыванием семян в популяции pOIL102+pOIL197 были равны 7,2%, 6,4% и 6,1%, соответственно (Таблицы 18-20). Важно, что максимальные наблюдаемые уровни TAG увеличивались в ходе развития растения от 2,7% (вегетативная стадия) до 3,5% (стадия трубчатого листа) и 4,3% (стадия зрелости с завязыванием семян) (Таблицы 18-20). По сравнению с данными, полученными для отдельных трансформаций с использованием трансгенов DGAT и WRI1, это является примером синергизма для совместной экспрессии трансгенов DGAT и WRI1 для увеличения накопления неполярных липидов в вегетативных тканях растений. Высокие уровни TAG и TFA были в большинстве случаев ассоциированы с значительным уменьшением содержания C18:39,12,15, которое было снижено приблизительно на 50% в линиях с наиболее высокими уроввнями TAG.
Тридцать шесть первичных трансформантов, содержащих как pOIL197 (pZmUbi:DGAT и pZmUbi:олеозин), так и pOIL103 (pZmPEPC:WRI1), анализировали по содержанию TFA и TAG, и составу жирных кислот на трех стадиях развития растения. Данные представлены в таблицах 21-23. Для некоторых растений с этой комбинацией генов показаны наивысшие уровни TFA и TAG, детектированные в этих сериях экспериментов. Наблюдаемые уровни TFA на вегетативной стадии, стадии трубчатого листа и на стадии зрелости с завязыванием семян в популяции pOIL103+pOIL197 составляли 8,3%, 8,3% и 4,5%, соответственно (Таблицы 21-23). Наблюдаемые уровни TAG на вегетативной стадии, стадии трубчатого листа и на стадии зрелости с завязыванием семян составляли 2,3%, 6,6% и 3,0%, соответственно (Таблицы 21-23). Следует отметить, что наивысшие уровни TAG (6,6%) и TFA (8,3%) среди всех трансгенных линий детектированы для события TX-03-31 на стадии трубчатого листа. В то время как C18:39,12,15, как правило, доминировали в фракции TFA, для составов TAG в этой популяции показана высокая степень изменчивости. Следует отметить, что для некоторых событий показано увеличение уровней пальмитиновой кислоты (C16:0) и/или линолеиновой кислоты (LA, C18:29,12) за счет ALA. Действительно, уровень ALA в содержании как TFA, так и TAG, был уменьшен ниже 40% для некоторых событий, менее чем 30% для избранных событий. Уровень ALA в TAG был менее чем 20% для некоторых избранных событий.
Растения, содержащие более высокие уровни TFA и TAG, размножали посредством отделения отростков и переноса их в грунт в новые горшки. Отростки образовывали новые корни и продолжали расти. При анализе образцов новых растений, наблюдали уровни TAG 8,3% при уровне TFA 9,3%.
В шестнадцати первичных трансформантах, содержащих как pOIL197 (pZmUbi:DGAT и pZmUbi:олеозин), так и pOIL104 (pSSU:WRI1), анализировали содержание TFA и TAG, и состав жирных кислот. В образцах листьев первичных трансформантов, содержащих области T-ДНК как pOIL197, так и pOIL104, собранных на вегетативной стадии роста, наблюдали 4,1% и 5,9% TFA (Таблица 24). Неожиданно, наивысшие уровни TFA, детектированные в этой популяции, сопровождались относительно низким содержанием TAG. Уровни TAG в трансгенных растениях pOIL104+pOIL197 на вегетативной стадии и стадии трубчатого листа достигали только 0,6% и 2,8%. Увеличенные уровни TAG были, как правило, ассоциированы с уменьшением содержания C18:39,12,15 и увеличением содержания как пальмитиновой кислоты, так и LA.
Уровни TFA и TAG во множестве независимых трансформированных растений схематически показаны на Фигуре 19.
Уровни экспрессии генов WRI1 и DGAT1 в ряде растений измеряли способом ОТ-ПЦР. Наблюдали, что растение TX-03-31, имеющее относительно высокое TTQ, имело наивысший уровень экспрессии DGAT среди тестированных растений. Сделано заключение, что высокие уровни экспрессии DGAT были благоприятными для увеличения уровня TAG, а также TTQ.
Возможно, наиболее удивительным и неожиданным заключением, выведенным из большого количества данных в этом Примере, являлся относительно высокий уровень TFA, сопровождающийся низкими уровнями TAG, за исключение немногих растений, таких как растение TX-03-31 (Таблица 22). То есть, хотя по существу происходил намного увеличенный синтез жирных кислот, большая часть увеличенного количества жирных кислот не проявлялась как TAG. Это заключение являлось полностью противоположным тому, что наблюдали для WRI1+DGAT трансгенных растений для Nicotiana, включая табак. Для количественной оценки этого в растениях сорго, соотношение уровней TAG и TFA рассчитывали для всех вышеупомянутых трансгенных популяций сорго (Таблицы 11-24). Параметр соотношения TAG/TFA (TTQ) рассчитывали как уровень TAG (%), деленный на уровень TFA (%), каждый как процент от сухой массы растительного материала (в этом случае. листа). Наблюдали, что для множества линий сорго, TTQ лежало в диапазоне от 0,01 до 0,6. Добавление одной или нескольких дополнительных генетических модификаций в растения, обеспечивающих уменьшение уровня SDP1, TGD или TST, или увеличение уровней одного или нескольких из полипептидов PDAT, PDCT или CPT увеличивало TTQ до более 0,6 для большой доли линий растений. В частности, уменьшение содержания TAG-липазы в растениях увеличивает TTQ до вплоть до 0,95.
Из-за больших различий абсолютных уровней TFA и TAG во многих трансгенных линиях, the авторы настоящего изобретения выбрали два события pOIL102+pOIL197 для количественной оценки главных классов нейтральных и полярных липидов, для определения типа липида, в котором присутствует высокий уровень жирных кислот. Типы липидов разделяли посредством TLC и оценивали количественно. На вегетативной стадии роста, TX-02-8 и TX-02-19 содержали 4,5% и 7,2% TFA, соответственно (Таблица 18). Содержание TAG только слабо увеличивалось в листьях TX-02-8, в то время как уровни фосфатидилхолина (PC, фосфолипида) и галактолипида MGDG были сравнимыми с отрицательным контролем. Для TX-02-19 показаны увеличенные уровни TAG, PC и MGDG, показывающие увеличение для классов как нейтральных, так и полярных липидов.
Более детальный анализ липидов проводили в растении TX-03-8 (на стадии трубчатого листа) и TX-03-28 (на вегетативной стадии) (Фигура 13). Дикий тип (при цветении) и трансформант с пустым вектором (на вегетативной стадии) служили в качестве контролей для сравнения. Несмотря на различия в возрасте растений на время сбора образцов, листья обоих трансгенных растений содержали увеличенные уровни TFA и общих полярных липидов. TX-03-28 содержало вплоть до 3,4% TAG на вегетативной стадии, в то время как уровни TAG в TX-03-8 были только слабо увеличены на стадии трубчатого листа. Для обеих трансгенных линий показано неожиданно большое увеличение количества галактолипидов MGDG и DGDG. Увеличения для различных классов полярных липидов, фосфолипидов PC, PG, PE, PA, PS, PI, являлись менее выраженными, но все еще значимыми (Фигура 13B). Дополнительное исследование посредством LC-MS выявило увеличенные уровни C18:0, C18:29,12 и C18:39,12,15 в фракции свободных жирных кислот для обеих трансгенных линий, что позволяет предполагать поток к PC посредством модификации ацила перед липолизом. Молекулярные виды DAG в тканях трансгенных листьев, содержание которых было увеличено, включали 34:2 (вероятно, C16:0/C18:29,12), 34:3 (вероятно, C16:0/C18:39,12,15), 36:4 (вероятно, C18:29,12/C18:29,12 и C18:19/C18:39,12,15) и 36:5 (вероятно,C18:29,12/C18:39,12,15). Обогащение фракции DAG полиненасыщенными жирными кислотами соответствовало составу TAG и позволяло предполагать происходящий из PC DAG в качестве предшественника для синтеза TAG. Подобные изменения молекулярных видов PC и PE наблюдали в обоих трансгенных растениях, в то время как виды PI в основном содержали C16:0 и C18 жирные кислоты. Молекулярные виды PG были в высокой степени обогащены C16:0, что отражает их пластидный синтез по прокариотическому пути. Галактолипиды в обеих трансгенных линиях в основном происходили из эукариотического липидного пути, т.е. были обогащены C18 жирными кислотами. Самым важным молекулярным видом MGDG являлся 36:6 (вероятно, C18:39,12,15/C18:39,12,15), служащий в качестве субстрата для синтеза DGDG 36:6. Второй по важности молекулярный вид DGDG в обеих трансгенных линиях, 34:3 (вероятно, C16:0/C18:39,12,15), также, вероятно, происходил из внепластидного источника. Содержание молекулярных видов TAG, состоящих из C16/C16/C18 (48:X), C16/C16/C18 (50:X) и C16/C18/C18 (52:x), было увеличено в тканях трансгенных листьев. Интересно, что содержание 54:8 (вероятно, C18:29,12/ C18:39,12,15/C18:39,12,15) и 54:9 (вероятно, C18:39,12,15/ C18:39,12,15/C18:39,12,15) было уменьшено по сравнению с отрицательным контролем. Совместно, эти результаты позволяют предполагать увеличенный поток ацильных цепей в TAG через PC в трансгенных линиях, в то время как биосинтез галактолипидов в основном происходит через эукариотический путь. Эти данные также привели авторов настоящего изобретения к пониманию того, что уменьшение активности TGD или увеличение содержания PDCT и/или CPT в растенииях, в дополнение к присутствующим трансгенам, может, вероятно, увеличивать уровни TFA и TAG.
Химерные конструкции ДНК для опосредованной Agrobacterium трансформации применяются для трансформации Zea mays (кукуруза), как описано в Gould et al., (1991). Вкратце, экспланты верхушек побегов культивируют с трансгенной Agrobacterium в течение двух суток перед перемещением на солевую среду МС, содержащую канамицин и карбенициллин. После нескольких кругов субкультуривирования, самопроизвольно формируются трансформированные побеги и корни, и экспланты переносят в грунт. Кроме того, конструкции применяют для трансформации Hordeum vulgare (ячмень) и Avena sativa (овес) с использованием способов трансформации, известных для указанных видов. Вкратце, в случае ячменя применяют культуры Agrobacterium для трансформации клеток в незрелых эмбрионах ячменя (сорт Golden Promise) в соответствии с опубликованными способами (Tingay et al., 1997; Bartlett et al., 2008) с некоторыми модификациями в том, что эмбрионы длиной от 1,5 до 2,5 мм выделяют из незрелых зерновок и удаляют эмбриональные оси. Полученные экспланты культивируют в течение 2-3 суток с трансгенной Agrobacterium, а затем культивируют в темноте в течение 4-6 недель на средах, содержащих тиментин и гигромицин для генерации эмбриогенного каллюса, после чего переносят в переходные среды в условиях слабой освещенности на две недели. Затем каллюсы переносят в регенерационные среды, чтобы позволить регенерацию побегов и корней, перед переносом регенерировавших ростков в грунт. Трансформированные растения получают и выращивают до стадии зрелости в теплице.
Таблица 11. Уровни TFA и TAG, состав жирных кислот и TTQ в отрицательном контроле дикого типа (WT) и в отрицательном контроле с пустым вектором (EV) в ходе различных стадий развития растения.
Вег.: Вегетативная; BL, Стадия роста с трубчатым листом; MSS, Стадия зрелости с завязыванием семян
Таблица 12. Уровни TFA и TAG, состав жирных кислот и TTQ в листьях сорго, трансформированных с использованием pOIL197 или pOIL136 (pZmUbi:DGAT; pZmUbi:олеозин) в ходе вегетативной стадии роста. Линии перечислены в порядке увеличения уровней TFA.
Таблица 13. Уровни TFA и TAG, состав жирных кислот и TTQ в листьях сорго, трансформированных с использованием pOIL197 или pOIL136 (pZmUbi:DGAT; pZmUbi:олеозин) в ходе стадии роста с трубчатым листом. Линии перечислены в порядке увеличения уровней TFA.
Таблица 14. Уровни TFA и TAG, состав жирных кислот и TTQ в листьях сорго, трансформированных с использованием pOIL197 или pOIL136 (pZmUbi:DGAT; pZmUbi:олеозин) в ходе стадии зрелости с завязыванием семян. Линии перечислены в порядке увеличения уровней TFA.
Таблица 15. Уровни TFA и TAG, состав жирных кислот и TTQ в листьях сорго, трансформированных с использованием pOIL102 (pZmUbi:WRI1) в ходе вегетативной стадии роста.
n3
Таблица 16. Уровни TFA и TAG, состав жирных кислот и TTQ в листьях сорго, трансформированных с использованием pOIL102 (pZmUbi:WRI1) в ходе стадии роста с трубчатым листом.
n3
Таблица 17. Уровни TFA и TAG, состав жирных кислот и TTQ в листьях сорго, трансформированных с использованием pOIL102 (pZmUbi:WRI1) в ходе стадии зрелости с завязыванием семян.
Таблица 18. Уровни TFA и TAG, состав жирных кислот и TTQ в листьях сорго, трансформированных с использованием pOIL102 (pZmUbi:WRI1) и pOIL197 (pZmUbi:DGAT и pZmUbi:олеозин) в ходе вегетативной стадии роста. Линии перечислены в порядке увеличения уровней TFA.
Таблица 19. Уровни TFA и TAG, состав жирных кислот и TTQ в листьях сорго, трансформированных с использованием pOIL102 (pZmUbi:WRI1) и pOIL197 (pZmUbi:DGAT и pZmUbi:олеозин) в ходе стадии роста с трубчатым листом. Линии перечислены в порядке увеличения уровней TFA.
n3
Таблица 20. Уровни TFA и TAG, состав жирных кислот и TTQ в листьях сорго, трансформированных с использованием pOIL102 (pZmUbi:WRI1) и pOIL197 (pZmUbi:DGAT и pZmUbi:олеозин) в ходе стадии зрелости с завязыванием семян.
Таблица 21. Уровни TFA и TAG, состав жирных кислот и TTQ в первичных трансформантах pOIL103+pOIL197 в условиях вегетативного роста.
n3
Таблица 22. Уровни TFA и TAG, состав жирных кислот и TTQ в первичных трансформантах pOIL103+pOIL197 на стадии трубчатого листа.
Таблица 23. Уровни TFA и TAG, состав жирных кислот и TTQ в первичных трансформантах pOIL103+pOIL197 на стадии зрелости с завязыванием семян.
Таблица 24. Уровни TFA и TAG, состав жирных кислот и TTQ в первичных трансформантах pOIL104 (pSSU:WRI1) +pOIL197 (pZmUbi:DGAT и pZmUbi:олеозин) в условиях вегетативного роста.
Химерные конструкции ДНК для опосредованной Agrobacterium трансформации применяются для трансформации Zea mays (кукуруза), как описано в Gould et al. (1991). Вкратце, экспланты верхушек побегов культивируют с трансгенной Agrobacterium в течение двух суток перед перемещением на солевую среду МС, содержащую канамицин и карбенициллин. После нескольких кругов субкультуривирования, самопроизвольно формируются трансформированные побеги и корни, и экспланты переносят в грунт. Кроме того, конструкции применяют для трансформации Hordeum vulgare (ячмень) и Avena sativa (овес) с использованием способов трансформации, известных для указанных видов. Вкратце, в случае ячменя применяют культуры Agrobacterium для трансформации клеток в незрелых эмбрионах ячменя (сорт Golden Promise) в соответствии с опубликованными способами (Tingay et al., 1997; Bartlett et al., 2008) с некоторыми модификациями в том, что эмбрионы длиной от 1,5 до 2,5 мм выделяют из незрелых зерновок и удаляют эмбриональные оси. Полученные экспланты культивируют в течение 2-3 суток с трансгенной Agrobacterium, а затем культивируют в темноте в течение 4-6 недель на средах, содержащих тиментин и гигромицин для генерации эмбриогенного каллюса, после чего переносят в переходные среды в условиях слабой освещенности на две недели. Затем каллюсы переносят в регенерационные среды, чтобы позволить регенерацию побегов и корней, перед переносом регенерировавших ростков в грунт. Трансформированные растения получают и выращивают до стадии зрелости в теплице.
Пример 6. Модифицированные признаки в двудольных растениях
Содержание масла в двудольных растениях вида Trifolium repens (клевер), бобовых, обычно применяемых в качестве пастбищной культуры, повышается при экспрессии комбинации генов WRI1, DGAT и олеозина в вегетативных частях. Конструкцию pJP3502 применяют для трансформации T. repens посредством опосредованной Agrobacterium трансформации (Larkin et al., 1996). Вкратце, генетическую конструкцию pJP3502 вводят в A. tumefaciens посредством стандартной процедуры электропорации. Бинарный вектор дополнительно содержит ген селективного маркера 35S:NptII в пределах T-ДНК. Трансформированные клетки Agrobacterium выращивают на твердых средах ЛБ с добавлением канамицина (50 мг/л) и рифампицина (25 мг/л) и инкубируют при 28°C в течение двух суток. Единичную колонию используют для инициации свежей культуры. После 48 часов энергичного культивирования, клетки Agrobacterium применяют для обработки семядоль T. repens (сорт. Haifa), вырезанных из набухшего семени, как описано Larkin et al. (1996). После совместного культивирования в течение трех суток экспланты обрабатывают 25 мг/л канамицина, чтобы отобрать трансформированные побеги, а затем переносят в среду для корнеобразования для формирования корней, перед переносом в грунт.
Шесть трансформированных растений, содержащих T-ДНК из pJP3502, были получены и перенесены в грунт в теплице. Повышенное содержание масла наблюдалось в ткани некоторых из растений, не относящейся к семенам, где одно растение продемонстрировало увеличение уровней TAG в листьях более чем в 4 раза. Такие растения пригодны в качестве корма для животных, например, посредством выращивания растений на пастбищах, что обеспечивает питание с повышенным содержанием энергии на единицу массы (плотность энергии) и приводит к увеличению темпов прироста у животных.
Дополнительно, конструкцию pJP3502 применяют для трансформации других бобовых растений, таких как люцерна посевная (Medicago sativa) и люцерна усеченная (Medicago truncatula), по способу Wright et al. (2006), с получением трансгенных растений с повышенным содержанием TAG в вегетативных частях. Трансгенные растения пригодны в качестве пастбищной культуры или в качестве сена или силоса как источника питания для животных, таких как крупный рогатый скот, овцы и лошади, обеспечивая повышенную плотность энергии в корме.
Пример 7. Модификация пластидной экспрессии GPAT
Повышенная экспрессия пластидной GPAT в клетках растений
Целый ряд экспериментов выполнялся для проверки гипотезы о том, что присутствие высокоактивного прокариотического пути 16:3 в растении (т.е. так называемом растении 16:3) обеспечивало бы значительно более низкие уровни TAG в вегетативных тканях при введении комбинации генов на pJP3502, относительно растений 18:3. Такие эксперименты описаны в следующих Примерах. Сначала, авторы настоящего изобретения оценивали, может ли повышенная экспрессия пластидной GPAT воспрепятствовать высокому уровню аккумуляции TAG в трансгенном N. benthamiana, увеличивая поток в прокариотическом пути.
Кодирующий участок для экспрессии пластидной GPAT Arabidopsis thaliana, ATS1 (Nishida et al., 1993), амплифицируют способом ОТ-ПЦР из общей РНК A. thaliana и клонируют в качестве фрагмента EcoRI-PstI в бинарный вектор экспрессии pJP3343 под контролем промотора 35S, с получением конститутивного вектора экспрессии pOIL098. Влияние повышенной экспрессии пластидной GPAT на генетическом фоне высокого содержания масла в листьях определяли посредством инфильтрации ткани листа с высоким содержанием масла химерным вектором pOIL098. Ткань листа с высоким содержанием масла генерируют совместной инфильтрацией бинарными векторами экспрессии WRI1 и DGAT (Пример 1) или инфильтрацией pOIL098 листьев растения Nicotiana, стабильно трансформированного T-ДНК из pJP3502 или другого вектора высокого содержания масла. Ожидается, что содержание масла будет снижено в инфильтрованных пятнах на листьях, соэкспрессирующих ген, кодирующий ATS1. Это определяют способом анализа TFA и TAG как доли от сухой массы образца. Дополнительно, это определяют посредством наблюдения за инкорпорацией меченного ацетата в жирные кислоты, продуцированные микросомами или листовыми лизатами, полученными из инфильтрованных пятен на листьях.
Накопление масла в Arabidopsis thaliana с мутантной пластидной GPAT
Мутант ats1 A. thaliana содержит разрушительную мутацию в гене, кодирующем пластидную GPAT, которая уменьшает активность пластидной GPAT до уровня только 3,8% от дикого типа (Kunst et al., 1988). Уровни аккумуляции TAG за пределами семян, по меньшей мере в листьях, стеблях и корнях, как в материнской A. thaliana, так и растении с мутантной ats1 оценивают и сравнивают. T-ДНК конструкции pJP3502 для повышенной экспрессии комбинации генов, кодирующих WRI1, DGAT и олеозин, вводят посредством трансформации в растения обоих генотипов. Комбинация генов в T-ДНК pJP3502 повышает синтез жирных кислот в обоих типах генетического фона растений. Однако, ожидается, что в присутствии мутантного ats1 накопление TAG в среднем будет существенно выше, чем в трансгенных растениях, полученных от генотипа дикого типа (материнский), за счет снижения активности пластидной GPAT и таким образом уменьшения потока жирных кислот в пластидный прокариотический путь. Соотношение C16:3 и C18:3 жирных кислот существенно уменьшается в листьях с мутантным ats1, как трансформированных, так и нетрансформированных.
Сайленсинг гена, кодирующего пластидную GPAT в клетках растений
В дополнение к генетической модификации растения введением мутации в ген, кодирующий пластидную GPAT, поток жирных кислот через прокариотический путь 16:3 может быть уменьшен и, таким образом, повышено содержание масла в вегетативных частях посредством сайленсинга пластидной GPAT. Это демонстрируется получением трансгенной кассеты, содержащей конститутивный или специфический для листьев промотор, экспрессирующий шпилечную РНК, соответствующую участку гена, кодирующего пластидную GPAT выбранного вида. В качестве примера, кассету экспрессии шпилечной РНКи получают с использованием фрагмента SalI-EcoRV последовательности кДНК пластидной GPAT A. thaliana размером 581 пара оснований (NM_179407, SEQ ID NO: 177). Кроме того, участок любого гена, кодирующего пластидную GPAT, последовательность которого в высокой степени идентична нуклеотидной последовательности NM_179407, можно применять для конструирования гена с целью экспрессии шпилечной РНК для сайленсинга эндогенного гена пластидной GPAT. Конструкция шпРНКи, содержащая фрагмент пластидной GPAT N. benthamiana размером 732 пары оснований (SEQ ID NO: 210), фланкированный уникальными сайтами SmaI и KasI, разработана с целью стабильной трансформации N. tabacum. Синтезированный фрагмент пластидной GPAT N. benthamiana субклонируют в сайты SmaI-KasI pJP3303, с получением pOIL113. Ожидается, что уменьшение удерживания жирных кислот в пластидах будет приводить к увеличению аккумуляции TAG, особенно при объединении с компонентом «Push», таким как повышенная экспрессия фактора транскрипции, такого как WRI1, или компонентом «Pull», таким как DGAT или PDAT и/или сниженная активность SDP1 или TGD.
Инактивация гена, кодирующего пластидную GPAT или, более того, любого гена может быть достигнута с использованием способов CRISPR/Cas9. Например, инактивация гена, кодирующего пластидную GPAT A. thaliana (номер доступа NM_179407) может осуществляться разрушением гена, опосредованным CRISPR/Cas9/короткой направляющей РНК (крРНК), с последующим мутагенезом посредством восстановления ДНК негомологичным соединением концов (NHEJ). Перед направленным расщеплением ДНК, Cas9 стимулирует разделение цепей ДНК и позволяет КНРНК гибридизоваться со специфической последовательностью длиной 20 нуклеотидов гена-мишени. Это помещает ДНК-мишень в активный сайт Cas9 в надлежащей ориентации относительно сайта связывания с PAM (тандемные гуанозиновые нуклеотиды). Такое расположение позволяет отдельным нуклеазным доменам Cas9 независимо расщеплять каждую цепь последовательности ДНК-мишени в точке, отстоящей на 3 нуклеотида в направлении против хода транскрипции относительно сайта PAM. Далее происходит восстановление двухцепочечного разрыва ДНК посредством подверженного ошибкам NHEJ, в ходе которого происходят делеции или инсерции нескольких нуклеотидов, что приводит к инактивации гена пластидной GPAT. Последовательности кнРНК, нацеленные на ген GPAT A. thaliana, идентифицированы и отобраны с использованием веб-инструмента CRISPRP (Xie et al., 2014). Последовательность-мишень длиной 20 нуклеотидов может быть любой последовательностью длиной 20 нуклеотидов в пределах гена-мишени, в том числе в пределах не кодирующих участков гена, таких как промотор или интрон, при условии, что она является специфической последовательностью в пределах генома. Последовательность может быть вставлена в бинарный вектор, содержащий кассету экспрессии CRISPR/Cas9/кнРНК и селективный маркер канамицина для растений (Jiang et al., 2013) и трансформирована в клетки растений посредством опосредованного Agrobacterium превращения. Можно осуществить скрининг трансгенных растений T1 на предмет мутаций в гене пластидной GPAT способом ПЦР амплификации и секвенирования ДНК.
Пример 8. Повышение экспрессии тиоэстеразы в клетках растений
Синтез жирных кислот de novo происходит в пластидах эукариотических клеток, где жирные кислоты синтезируются при связывании с ацилпереносящим белком в форме конъюгатов ацил-ACP. После удлинения цепи до ацильных групп C16:0 и C18:0, а затем введения кратной связи в C18:1 во время связывания с ACP, жирные кислоты отщепляются от ACP тиоэстеразами и входят в эукариотический путь посредством экспорта из пластид и транспорта в ER, где они принимают участие в биогенезе мембран и запасных липидов. В хлоропластах процесс экспорта включает две стадии: во-первых, ацильные цепи высвобождаются в форме свободных жирных кислот по действием ферментов тиоэстераз (тиоэстераза жирного ацила; FAT), во-вторых, введение в реакцию с CoA с образованием эфиров ацил-CoA, что катализируется длинноцепочечными ацил-CoA-синтетазами (LACS). A. thaliana содержит 3 тиоэстеразы жирного ацила, которые могут отличаться на основании их специфичности в отношении ацильной цепи. FATA1 и FATA2 преимущественно гидролизуют ненасыщенные ацил-ACP, в то время как насыщенные цепи ацил-ACP обычно расщепляются FATB.
С целью изучения влияния соэкспрессии тиоэстеразы и генов, кодирующих полипептиды WRI1 и/или DGAT, на содержание общих жирных кислот, содержание TAG и состав жирных кислот получают химерные гены для каждой из трех тиоэстераз A. thaliana посредством инсерции кодирующих участков в бинарный вектор экспрессии pJP3343 для временной экспрессии в клетках листьев N. benthamiana промотором 35S. Кодирующие белок участки FATA1 A. thaliana (номер доступа NP_189147.1, SEQ ID NO: 193) и FATA2 (номер доступа NP_193041.1, SEQ ID NO: 194) амплифицируют из кДНК стручков с использованием праймеров, содержащих сайты EcoRI и PstI, и далее клонируют в pJP3343 с использованием таких же рестрикционных сайтов. Полученные векторы экспрессии обозначены pOIL079 и pOIL080, соответственно. Кодирующий белок участок гена FATB A. thaliana (номер доступа NP_172327.1, SEQ ID NO: 195) амплифицируют с использованием праймеров, содержащих фланкированные сайты NotI и SacI, и клонируют в соответствующие рестрикционные сайты pJP3343, с получением pOIL081. Конструкциями pOIL079, pOIL080 и pOIL081 инфильтруют ткань листа N. benthamiana, отдельно или в комбинации с конструкциями, содержащими гены. фактора транскрипции WRI1 A. thaliana (AtWRI1) (pJP3414) и/или DGAT1 ацилтрансферазы (AtDGAT1) (pJP3352). Для сравнения, химерные гены, кодирующие FatB1 Cocos nucifera (CnFATB1) (pJP3630), FatB2 C. nucifera (CnFATB2) (pJP3629) вводят в ткань листа N. benthamiana параллельно с тиоэстеразами Arabidopsis, чтобы сравнить влияние полипептидов FatB, обладающих специфичностью в отношении MCFA, с тиоэстеразами Arabidopsis, не обладающими специфичностью в отношении MCFA. Все эксперименты с инфильтрацией включают химерный ген для экспрессии супрессора сайленсинга p19, как описано в Примере 1. Отрицательный контроль инфильтруют только T-ДНК p19.
Наблюдали синергический эффект для экспрессии тиоэстеразы и повышенной экспрессии WRI1 и/или DGAT на уровни TAG в листьях N. benthamiana. Экспрессия генов тиоэстеразы без генов WRI1 или DGAT существенно повышает уровни TAG свыше низкого уровня в отрицательном контроле (только p19). Например, экспрессия тиоэстеразы FATB2 кокоса приводила к увеличению уровней TAG в листьях в 8,2 раза, по сравнению с отрицательным контролем. Соэкспрессия фактора транскрипции WRI1 A. thaliana с каждой из тиоэстераз дополнительно повышала уровни TAG, по сравнению с контролем AtWRI1. Соэкспрессия каждой из тиоэстераз CnFATB1 и CnFATB2 кокоса с WRI1 приводила к более высоким уровням TAG, чем каждой из трех тиоэстераз A. thaliana с WRI1. Интересно, что противоположное наблюдалось, если ацилтрансфераза DGAT1 A. thaliana соэкспрессировалась в комбинации с тиоэстеразой и WRI1. Это наводит на мысль о лучшей специфичности в отношении ацильной цепи тиоэстераз A. thaliana и ацилтрансферазы DGAT1 A. thaliana, что приводит к увеличенному потоку ацильных цепей от ацил-ACP в TAG. Кроме того, не-MCFA тиоэстеразы были значительно более эффективны с точки зрения увеличения процента олеиновой кислоты в общем содержании жирных кислот в листьях. Соэкспрессия AtWRI1, AtDGAT1 и AtFATA2 приводила к наиболее высокому уровню TAG в листьях, обеспечивая уровень, который более чем в 1,6 раза превышал наблюдаемый при соэкспрессии AtWRI1 и AtDGAT1 без тиоэстеразы. Указанные эксперименты подтвердили синергическое увеличение синтезе и аккумуляции масла, при соэкспрессии WRI1 и DGAT, а также продемонстрировали дальнейшее синергическое увеличение при добавлении тиоэстеразы к комбинации.
Три различных бинарных вектора экспрессии сконструированы для оценки влияния соэкспрессии генов, кодирующих WRI1, DGAT1 и FATA, на уровни TAG и состав жирных кислот в листьях стабильно трансформированного N. tabacum. Вектор pOIL121 содержит ген SSU::AtWRI1 для экспрессии AtWRI1 промотором SSU, гена 35S::AtDGAT1 для экспрессии AtDGAT промотором 35S и гена enTCUP2::AtFATA2 для экспрессии AtFATA2 промотором enTCUP2, который является конститутивным промотором. Указанные генетические конструкции получают из pOIL38, сначала расщепляя ДНК с помощью NotI для удаления гена, кодирующего олеозин S. indicum. Кодирующий белок участок гена FATA2 A. thaliana амплифицируют и фланкируют сайтами NotI с использованием ДНК pOIL80 в качестве шаблона. Далее этот фрагмент вставляют в сайт NotI pOIL38. Затем pOIL121 служит материнским вектором для pOIL122, который содержит дополнительную кассету шпилечной РНК enTCUP2::SDP1 для опосредованного РНКи сайленсинга эндогенного гена SDP1 в трансгенных растениях. Чтобы этого достичь, полноразмерную кассету шпильки SDP1 N. benthamiana выделяют из pOIL51 (Пример 2) в виде фрагмента SfoI-SmaI и клонируют в сайт SfoI pOIL121, с получением pOIL122 (Фигура 14). Третий вектор, pOIL123, содержащий гены SSU::WRI1 и 35S::DGAT1 и ген шпилечной РНК enTCUP2::SDP1 получают подобным образом, клонируя кассету шпилечной РНК enTCUP2::SDP1 в виде фрагмента SfoI-SmaI в сайт SfoI pOIL36.
Вкратце, векторы содержали комбинации генов:
pOIL121: SSU::AtWRI1, 35S::AtDGAT1, enTCUP2::AtFATA2.
pOIL122: SSU::AtWRI1, 35S::AtDGAT1, enTCUP2::AtFATA2, enTCUP2::шпилька SDP1.
pOIL123: SSU::AtWRI1, 35S::AtDGAT1, enTCUP2::шпилька SDP1.
Каждую из трех конструкций применяют для получения трансформированных растений N. tabacum (сорт Wi38) посредством опосредованной Agrobacterium трансформации. Каждое из соэкспрессии тиоэстеразы FATA2 A. thaliana или сайленсинга эндогенной TAG-липазы SDP1 в комбинации с экспрессией AtWRI1 и AtDGAT1 приводило к дальнейшему увеличению уровней TAG, по сравнению с экспрессией AtWRI1 и AtDGAT1 в отсутствие как гена тиоэстеразы, так и гена сайленсинга SDP1. Наиболее высокий выход TAG получают с использованием pOIL122 посредством сочетанного действия всех четырех химерных генов.
Необходимо отметить, что N. benthamiana является растением 18:3. Такие же конструкции pOIL079, pOIL080 и pOIL081 применяются для трансформации A. thaliana, растения 16:3.
Авторы настоящего изобретения разработали модель, повышающую пластидный экспорт жирных кислот, например, снижения аккумуляции ацил-ACP в пластидах посредством повышения активности тиоэстеразы жирного ацила, таким образом, повышая биосинтеза жирных кислот в результате снижения ингибирования ацетил-CoA карбоксилазы (ACCазы) по механизму обратной связи (Andre et al., 2012; Moreno-Perez et al., 2012). Повышенная экспрессия тиоэстеразы увеличивает экспорт ацильных цепей из пластид в ER, таким образом, предлагая эффективную связь между так называемыми «Push» и «Pull» стратегиями метаболического конструирования.
Пример 9. Эффект различных полипептидов факторов транскрипции на признаки растений
В экспериментах с WRI1 и DGAT, о которых сообщалось ранее (Vanhercke et al., 2013), применяли синтетический ген, кодирующий AtWR I1 A. thaliana (номер доступа AAP80382.1), и синтетический ген, кодирующий AtDGAT1, также из A. thaliana (номер доступа AAF19262; SEQ ID NO: 1). Для сравнения других полипептидов WRI с AtWRI1 на предмет их способности объединяться с DGAT для повышения содержания масла, другие последовательности, кодирующие WRI, были идентифицированы и использованы при генерации конструкций для экспрессии в листьях N. benthamiana. Нуклеотидные последовательности, кодирующие факторы транскрипции A. thaliana WRI3 (номер доступа AAM91814.1, SEQ ID NO: 196) и WRI4 (номер доступа NP_178088.2, SEQ ID NO: 197) (To et al., 2012), синтезированы и вставлены в виде фрагментов EcoRI в pJP3343 под контролем промотора 35S. Результирующие бинарные векторы экспрессии обозначены pOIL027 и pOIL028, соответственно. Кодирующую последовательность для WRI1 (AsWRI1, SEQ ID NO: 198) овса (Avena sativa) амплифицируют способом ПЦР из вектора, предложенного проф. Sten Stymne (Шведский университет сельскохозяйственных наук), с использованием фланкирующих праймеров, содержащих дополнительные сайты EcoRI. Амплифицированный фрагмент вставляют в pJP3343 с получением pOIL055. Последовательность-кандидат WRI1 из S. bicolor (номер доступа XP_002450194.1, SEQ ID NO: 199) идентифицирована поиском BLASTp на сервере Национального центра биотехнологической информации (НЦБИ) с использованием аминокислотной последовательности WRI1 Zea mays (номер доступа NP_001137064.1, SEQ ID NO: 200) в качестве запроса. Кодирующий белок участок гена WRI1S. bicolor (SbWRI1) синтезирован и вставлен в виде фрагмента EcoRI в pJP3343, с получением pOIL056. Ген-кандидат, кодирующий WRI1, идентифицирован из транскриптомы китайского сального дерева (Triadica sebifera; TsWRI1, SEQ ID NO: 201) (Uday et al., подана). Кодирующий белок участок синтезирован и вставлен в виде фрагмента EcoRI в pJP3343, с получением pOIL070, Бинарные векторы pJP3414 и pJP3352, содержащие кодирующие последовательности для экспрессии. полипептидов WRI1 и DGAT1 A. thaliana являются такими, как описано Vanhercke et al. (2013).
Плазмиды, содержащие последовательности, кодирующие различные WRI, вводят в листовую ткань N. benthamiana для временной экспрессии с использованием гена, кодирующего белок вирусного супрессора p19 во всех процедурах инокуляции, как описано в Примере 1. Гены, кодирующие полипептиды WRI, оценивают отдельно или в комбинации с геном ацилтрансферазы DGAT1, где задача последнего состоит в обеспечении более высокого биосинтеза и аккумуляции TAG. Положительным контролем в данном эксперименте служит комбинация генов, кодирующих фактор транскрипции WRI1 A. thaliana и AtDGAT1. Все процедуры инфильтрации осуществляют в трех экземплярах с использованием трех различных растений, и уровни TAG анализируют, как описано в Примере 1. Экспрессия большинства индивидуальных полипептидов WRI в отсутствие экзогенно введенного DGAT1 приводит к повышенным, но все еще низким уровням TAG (< 0,23%, в пересчете на сухую массу) в инфильтрованных зонах листа, по сравнению с контролем, который содержал только конструкцию p19 (Фигура 15). Исключение составляет TsWRI1, который, сам по себе, по-видимому, не повышает значительно уровней TAG. Кроме того, отличия в уровнях TAG, полученных при экспрессии различных факторов транскрипции WRI, сами по себе не являются большими. Как AsWRI1, так и SbWRI1 давали уровни TAG, подобные собственно AtWRI1. Анализ состава жирных кислот TAG выявляет только незначительные изменения, за исключением повышенных уровней C18:1Δ9 при экспрессии AtWRI3 в инфильтрованных листовых тканях (Таблица 25).
Таблица 25. Состав жирных кислот TAG в образцах листьев N. benthamiana, инфильтрованных различными химерными генами для экспрессии WRI (n=3).
Все образцы дополнительно инфильтрованы конструкцией P19. Образцы TAG дополнительно содержат 0,1-0,4% C14:0; 0,5-1,2% C16:3 и; 0,1-0,7% C18:1Δ11.
В противоположность этому, отличия в выходах TAG при экспрессии различных полипептидов WRI являются более выраженными при соэкспрессии с ацилтрансферазой AtDGAT1. Это снова демонстрирует синергический эффект соэкспрессии WRI1 и DGAT на биосинтез TAG в инфильтрованной ткани листа N. benthamiana, как сообщается Vanhercke et al. (2013). Промежуточные уровни TAG наблюдаются при соэкспрессии DGAT1 с векторами экспрессии AtWRI3, AtWRI4 и TsWRI1, в то время как уровни, полученные с AsWRI1 и AtWRI1, значительно ниже. В качестве результата, который не мог быть предсказан заранее, наиболее высокие выходы TAG получены при соэкспрессии DGAT с SbWRI1, даже если эксперимент проводят на клетках двудольных. Анализ состава жирных кислот обнаруживает повышенные уровни TAG C18:1Δ 9 и сниженные уровни C18:3Δ 9,12,15 (ALA) в случае SbWRI1, AsWRI1 и положительного контроля AtWRI1. Однако, в отличие от AtWRI1, экспрессия AsWRI1 и SbWRI1 в обоих случаях продемонстрировала повышенные уровни C16:0, по сравнению с отрицательным контролем p19. Интересно, что инфильтрованные AtWRI3 образцы листьев продемонстрировали отличный профиль TAG, обогащенный C18:1Δ 9, в то время как C16:0 и ALA изменения только слегка коснулись.
Данный эксперимент демонстрирует, что фактор транскрипции WRI1 S. bicolor, SbWRI1, превосходит AtWRI1 при соэкспрессии с DGAT с точки зрения повышения уровней TAG в вегетативных частях растения. Кроме того, авторами изобретения сделан вывод о том, что фактор транскрипции, например, WRI1, из однодольного растения мог бы надлежащим образом функционировать в клетке двудольного растения, и, в действительности, даже мог бы обладать превосходящей активность, по сравнению с соответствующем фактором транскрипции двудольного растения. Кроме того, фактор транскрипции двудольного растения мог бы надлежащим образом функционировать в клетке однодольного растения.
Использование других факторов транскрипции
Получены генетические конструкции для экспрессии каждого из 14 различных факторов транскрипции в клетках растений, с целью оценки их способности функционировать в плане повышения уровней TAG в комбинации с другими генами, принимающими участие в биосинтезе и аккумуляции TAG. Указанные факторы транскрипции были кандидатами в качестве альтернатив WRI1 или для добавления к комбинациям, содержащим один или более из факторов транскрипции WRI1, LEC1 и LEC2, для применения в клетках растений, особенно в вегетативных частях растения. Их выбор в значительной степени основан на сообщениях об их участии в эмбриогенезе (обзор в Baud и Lepiniec (2010), а также Ikeda et al. (2006)), подобно LEC2. Таким образом, проведены эксперименты для оценки их функции с использованием системы экспрессии N. benthamiana (Пример 1), как указано ниже.
Нуклеотидные последовательности участков, кодирующих белок для следующих факторов транскрипции кодон-оптимизированы для экспрессии в N. benthamiana и N. tabacum, синтезированы и субклонированы в виде фрагментов NotI-SacI в соответствующие сайты pJP3343: A. thaliana FUS3 (pOIL164) (Luerssen et al., 1998; номер доступа AAC35247; SEQ ID NO:160), A. thaliana LEC1L (pOIL165) (Kwong et al. 2003; номер доступа AAN15924; SEQ ID NO:157), A. thaliana LEC1 (pOIL166) (Lotan et al., 1998; номер доступа AAC39488; SEQ ID NO:149), G. max MYB73 (pOIL167) (Liu et al., 2014; номер доступа ABH02868; SEQ ID NO:212), A. thaliana bZIP53 (pOIL168) (Alonso et al., 2009; номер доступа AAM14360; SEQ ID NO:213), A. thaliana AGL15 (pOIL169) (Zheng et al., 2009; номер доступа NP_196883; SEQ ID NO:214), A. thaliana MYB118 (номер доступа AAS58517; pOIL170; SEQ ID NO:215), MYB115 (Wang et al., 2002; номер доступа AAS10103; pOIL171; SEQ ID NO:216), A. thaliana TANMEI (pOIL172) (Yamagishi et al., 2005; номер доступа BAE44475; SEQ ID NO:217), A. thaliana WUS (pOIL173) (Laux et al., 1996; номер доступа NP_565429; SEQ ID NO:218), A. thaliana BBM (pOIL174) (Boutilier et al., 2002; номер доступа AAM33893, SEQ ID NO:145), B. napus GFR2a1 (номер доступа AFB74090; pOIL177; SEQ ID NO:219) и GFR2a2 (номер доступа AFB74089; pOIL178; SEQ ID NO:220) (Liu et al. (2012)). Кроме того, кодон-оптимизированная версия фактора транскрипции PHR1 A. thaliana, принимающего участие в адаптации к условиям фосфатного голодания при высокой освещенности, подобным образом субклонирована в pJP3343 (pOIL189) (Nilsson et al. (2012); номер доступа AAN72198; SEQ ID NO: 221). Указанные факторы транскрипции кратко описаны в Таблице 26.
В качестве скринингового теста для определения функции указанных факторов транскрипции, проводили совместную инфильтрацию в клетки листьев N. benthamiana, как описано в Примере 1, генетических конструкций и гена, кодирующего DGAT1, с геном, кодирующим WRI1, или без него. Общее содержание липидов и состав жирных кислот в клетках листьев анализировали через 5 суток после инфильтрации. Среди различных тестированных эмбриогенных факторов транскрипции, только сверхэкспрессия FUS3 приводила к значимо увеличенным уровням TAG в тканях листьев N. benthamiana по сравнению с контрольными инфильтрациями DGAT и DGAT1+WRI1 (Таблица 27).
Таблица 26. Дополнительные факторы транскрипции и генетические конструкции для их экспрессии
Таблица 27. Уровень TAG (% сухой массы листа) и профиль жирных кислот в листьях N. benthamiana после инфильтрации.
Для стабильной трансформации растений с использованием генов, кодирующих альтернативные факторы транскрипции, получены следующие бинарные конструкции. Гены для экспрессии факторов транскрипции используют промотор SSU или промотор SAG12. Показано, что повышенная экспрессия эмбриогенных факторов транскрипции, таких как LEC1 и LEC2, индуцирует различные плеотропные эффекты, нежелательные в настоящем контексте, включая соматический эмбриогенез (Feeney et al. (2012); Santos-Mendoza et al. (2005); Stone et al. (2008); Stone et al. (2001); Shen et al. (2010)). Для минимизации возможного отрицательного влияния на развитие растений и выход биомассы, специфичные для ткани или стадии развития промоторы являются предпочтительнее конститутивных промоторов с точки зрения управления эктопической экспрессией основных регуляторов эмбриогенеза.
Пример 10. Специфическая для стебля экспрессия гена, кодирующего фактор транскрипции
Листья растений N. tabacum, экспрессирующих трансгены, которые кодируют WRI1, DGAT и олеозин, на стадии завязывания семян содержат приблизительно 16% TAG. Однако, уровни TAG намного ниже в стеблях (1%) и корнях (1,4%) растений (Vanhercke et al., 2014). Авторами изобретения было оценено, являются ли более низкие уровни TAG в стеблях и корнях результатом слабой промоторной активности промотора SSU Rubisco, применяемого для экспрессии гена, кодирующего WRI1 в трансгенных растениях. Трансген DGAT в T-ДНК pJP3502 экспрессируется промотором CaMV35S, который более активно экспрессируется в стеблях и корнях, и таким образом он вряд ли является ограничивающим фактором для аккумуляции TAG в стеблях и корнях.
В попытке увеличить биосинтез TAG в ткани стебля, разработана конструкция, в которой ген, кодирующий WRI1, размещен под контролем. промотора SDP1 A. thaliana. Синтезирован синтетический фрагмент ДНК размером 3,156 тыс. пар оснований, содержащий промотор SDP1 A. thaliana размером 1,5 тыс. пар оснований (SEQ ID NO: 175) (Kelly et al., 2013), и далее участок, кодирующий полипептид WRI1 A. thaliana, и участок терминатора/полиаденилирования лектина G. max. Указанный фрагмент вставлен между сайтами SacI и NotI pJP3303. Результирующий вектор обозначен как pOIL050, и в дальнейшем применим для трансформации клеток растений N. tabacum, гомозиготных по T-ДНК из pJP3502 посредством опосредованной Agrobacterium трансформации. Трансгенные растения отобраны на основе устойчивости к гигромицину, где всего 86 независимых трансгенных растений выращены в теплице до стадии зрелости. Получены образцы ткани трансгенного листа и стебля на стадии завязывания семян, которые содержат более высокие уровни TAG, чем материнские растения N. tabacum, трансформированные pJP3502.
Пример 11. Влияние экспрессии белка масляных телец на признаки растений
Растения N. tabacum, трансформированные T-ДНК pJP3502 и экспрессирующие трансгены, которые кодируют WRI1, DGAT1 A. thaliana и олеозин S. indicum, содержат повышенные уровни TAG в вегетативных тканях. Как проиллюстрировано в Примере 2 выше, при сайленсинге эндогенного гена, кодирующего липазу TAG SDP1, в таких растениях, уровни TAG в листьях дополнительно повышаются, что указало авторам изобретения на тот факт, что происходит значительный оборот TAG в растениях, которые сохранили активность SDP1. Таким образом, был определен уровень экспрессии трансгенов в растениях. Хотя гибридизация способом Нозерн-блоттинга подтвердила выраженную экспрессию WRI1 и DGAT1 и некоторую экспрессию мРНК олеозина, анализ экспрессии способом цифровой ПЦР и кОТ-ПЦР обнаружил только очень низкие уровни транскриптов олеозина. Анализ экспрессии выявил, что ген, кодирующий олеозин, экспрессируется слабо, по сравнению с трансгенами WRI1 и DGAT1. На основании указанных экспериментов, авторами изобретения был сделан вывод о том, что масляные тельца в ткани листа не были полностью защищены от расщепления TAG из-за ненадлежащей продукции белка олеозина, кодируемого T-ДНК в pJP3502. Для повышения стабильности аккумуляции TAG в процессе развития растения, были разработаны несколько модификаций pJP3502, в которых ген олеозина был заменен. Указанные модифицированные конструкции были такими, как указано ниже.
1. pJP3502 содержит ген (в SEQ ID NO: 176 приведена комплементарная ему последовательность), кодирующий олеозин S. indicum, который слабо экспрессировался. Данный ген содержит внутренний интрон UBQ10, который может снижать уровень экспрессии. Для проверки этого, синтетический фрагмент ДНК размером 502 пары оснований, содержащий ген олеозина S. indicum и не содержащий внутреннего интрона UBQ10, был синтезирован и вставлен в pJP3502 в виде фрагмента NotI, заменяя в pJP3502 ген олеозина, содержащий интрон. Результирующая плазмида обозначена как pOIL040.
2. Промотор маленькой субъединицы (SSU) Rubisco, управляющий экспрессией гена олеозина в pJP3502, был заменен конститутивным промотором enTCUP2. На данный момент, фрагмент размером 2321 пар оснований, содержащий промотор enTCUP2, кодирующий белок олеозин участок, участок терминатора/полиаденилирования лектина G. max и первые 643 пары оснований в направлении по ходу транскрипции промотора SSU, управляющего экспрессией wri1, синтезирован и субклонирован в сайты AscI и SpeI pJP3502, с получением pOIL038.
3. Подобной стратегией следовали для экспрессии сконструированной версии гена олеозина S. indicum, содержащего 6 введенных остатков цистеина (o3-3) под контролем промотора enTCUP2 (Winichayakul et al., 2013). Фрагмент размером 2298 пар оснований, содержащий промотор enTCUP2, кодирующий белок олеозин участок o3-3, участок терминатора/полиаденилирования лектина G. max и первые 643 пары оснований в направлении по ходу транскрипции промотора SSU, управляющего экспрессией wri1, синтезирован и субклонирован в сайты AscI и SpeI pJP3502, с получением pOIL037.
4. Сайты NotI, фланкирующие ген олеозина S. indicum в pJP3502, применяются для обмена кодирующего белок участка на участок, кодирующий олеозин 3 арахиса (номер доступа AAU21501.1) (Parthibane et al., 2012a; Parthibane et al., 2012b). Фрагмент размером 528 пар оснований, содержащий ген олеозина 3, фланкированный сайтами NotI, синтезирован и субклонирован в соответствующий сайт pJP3502. Результирующий вектор обозначен как pOIL041
5. Дополнительно, фланкированный NotI фрагмент размером 1077 пар оснований, который содержит ген, кодирующий стеролеозин A. thaliana (Arab-1) (номер доступа AAM10215.1) (Jolivet et al., 2014), синтезирован и субклонирован в сайт NotI pJP3502, с получением pOIL043.
6. Поверхностный белок липидных капелек Nannochloropsis oceanic (ПБЛК) (номер доступа AFB75402.1) (Vieler et al., 2012) синтезирован в виде NotI-фланкированного фрагмента размером 504 пары оснований и субклонирован в сайт NotI pJP3502, с получением pOIL044.
7. И наконец, калеозин A. thaliana (CLO3) (номер доступа O22788.1) (Shimada et al., 2014) синтезирован в виде фланкированного NotI фрагмента размером 612 пар оснований и субклонирован в pJP3502, с получением pOIL042.
Каждую из этих конструкций вводили в клетки листьев N. benthamiana, как описано в Примере 1. Временная экспрессия как pJP3502, так и pOIL040 в ткани листа N. benthamiana приводит к повышенным уровням TAG и подобным изменениям профиля жирных кислот в TAG, однако pOIL040 повышает уровень TAG в большей степени (1,3% по сравнению с 0,9%). Каждая из конструкций pOIL037, pOIL038, pOIL041, pOIL042 и pOIL043 применим для стабильной трансформации растений N. tabacum (сорт W38) с помощью опосредованных Agrobacterium способов. Трансгенные растения отбирают на основании устойчивости к канамицину и выращивают в теплице до состояния зрелости. На различных стадиях развития растения отбирают образцы трансгенной ткани листа, которые содержат повышенные уровни TAG, по сравнению с N. tabacum дикого типа и растениями N. tabacum, трансформированными pJP3502.
Клонирование и характеристика полипептидов LDAP из Sapium sebiferum
Олеозины не экспрессируются с высокими уровнями в несеменных тканях растения, накапливающих масло, таких как мезокарп оливы, масличной пальмы и авокадо (Murphy, 2012). Вместо этого, в данных тканях идентифицированы белки, связанные с липидными капельками (LDAP), которые могут играть роль, подобную роли олеозина в тканях семени (Horn et al., 2013). Таким образом, авторы настоящего изобретения рассмотрели возможность того, что олеозин не является оптимальным упаковочным белком для защиты аккумулированного масла от TAG-липазы или другого влияния цитозольных ферментов в вегетативных тканях растений. Таким образом, полипептиды LDAP были идентифицированы и оценены на предмет повышения аккумуляции TAG, как указано ниже.
Плод китайского сального дерева, Sapium sebiferum, члена семейства Euphorbiaceae, представляет особый интерес для авторов изобретения, поскольку он содержит богатую маслом ткань за пределами семени. Недавнее исследование (Divi et al., подано для публикации) показало, что указанная маслянистая ткань под названием жировой слой может происходить из мезокарпа плода растения. Таким образом, авторами изобретения подвергнута сомнению транскриптома S. sebiferum из-за последовательностей LDAP. Сравнительный анализ экспрессированных генов в тканях оболочки фрукта и семени выявил группу из трех ранее идентифицированных генов LDAP, которые экспрессировались с высокими уровнями в жировом слое.
Нуклеотидные последовательности, кодирующие три LDAP, получены способом ОТ-ПЦР с использованием РНК, происходящих из жировой ткани, и трех пар праймеров. Последовательности праймеров основаны на последовательностях ДНК, фланкирующих полный кодирующий участок для каждого из трех генов. Последовательности праймера: для LDAP1, 5'-TTTTAACGATATCCGCTAAAGG-3' (SEQ ID NO: 236) и 5'-AATGAATGAACAAGAATTAAGTC-3' (SEQ ID NO: 237) AT-3'; LDAP2, 5'-CTTTTCTCACACCGTATCTCCG-3' (SEQ ID NO: 238) и 5'-AGCATGATATA CTTGTCGAGAAAGC-3' (SEQ ID NO: 239); LDAP3, 5'-GCGACAGTGTAGCGTTTT-3' (SEQ ID NO: 240) и 5'-ATACATAAAATGAAAACTATTGTGC-3' (SEQ ID NO: 241).
Анализ транскриптома S. sebiferum обнаружил многочисленные ортологи для каждого из генов LDAP, в том числе восемь LDAP1, шесть LDAP2 и шесть LDAP3 генов, с менее чем 10% дивергенцией последовательностей в пределах каждого семейства гена. Предполагаемые последовательности пептида выровнены, и филогенетическое дерево сконструировано с использованием программного обеспечения Genious (Фигура 16), вместе с гомологами LDAP из других видов растений, включая два из авокадо (Pam), один из масличной пальмы, один из Parthenium argentatum (Par), два из Arabidopsis (Ath), пять из Taraxacum brevicorniculatum (Tbr), три из Hevea brasiliensis (Hbr), как проиллюстрировано на Фигура 16. Филогенетическое дерево выявило, что SsLDAP3 разделяет большую степень идентичности аминокислотных последовательностей с полипептидами LDAP1 и LDAP2 из авокадо и LDAP из масличной пальмы, в то время как полипептиды SsLDAP1 и SsLDAP2 были более дивергентными.
Генетические конструкции для повышенной экспрессии LDAP
Для оценки функции LDAP из S. sebifera получены векторы экспрессии для экспрессии каждого из указанных полипептидов под контролем промотора 35S в клетках листа. Полноразмерные последовательности кДНК SsLDAP вставляют в вектор назначения pDONR207 посредством реакций рекомбинации, заменяя участки CcdB и Cm(R) вектора назначения фрагментами кДНК SsLDAP. Последующее подтверждение способом анализа рестрикционного расщепления и секвенирования ДНК, конструкции вводят в штамм AGL1 Agrobacterium tumefaciens и применяют для временной экспрессии в клетках листьев N. benthamiana и стабильной трансформации N. tabacum.
Экспрессия каждого из трех генов SsLDAP под транскрипциональным контролем промотора 35S в листьях N. benthamiana в комбинации с экспрессией 35S::AtDGAT1 и 35S::AtWRI1 дает значительно более высокие уровни аккумуляции TAG, по сравнению с клетками, инфильтрованными генами 35S::AtDGAT1 и 35S::AtWRI1 без конструкции LDAP. Уровень TAG повышается приблизительно в 2 раза, по сравнению с уровнем TAG в контрольных клетках. В клетках, получающих комбинацию генов, наблюдается значительное увеличение уровня α-линоленовой кислоты (ALA) и снижение уровня насыщенных жирных кислот, относительно контрольных клеток.
Солокализация слитых с YFP полипептидов LDAP с капельками липида в клетках листа
С целью характеристики SsLDAP in vivo и наблюдения за их поведением в динамике, получены экспрессионные конструкции для экспрессии химерных полипептидов, состоящих из полипептидов LDAP, слитых с желтым флуоресцентным белком (YFP). Для каждого химерного полипептида, YFP был слит в рамке с C-концом SsLDAP. Полная открытая рамка считывания каждого из трех генов LDAP, без стоп-кодона на 3' конце, слита с последовательностью YFP и химерными генами, вставленными в pDONR207. После подтверждения полученных конструкций способом рестрикционного расщепления и секвенирования ДНК, в конструкции вводят в штамм AGL1 A. tumefaciens и применяют для временной экспрессии в клетках листа N. benthamiana и стабильной трансформации N. tabacum. Через три дня после инфильтрации клеток листа конструкциями LDAP-YFP, листовые диски из инфильтрованных областей окрашивают Красным Нилом, который положительно окрашивает липидные капельки, и визуализируют под однофокусным микроскопом, чтобы обнаружить красное окрашивание (капельки липида) и флуоресценцию полипептида YFP. Наблюдается солокализация LDAP-YFP с липидными капельками, указывая на связь LDAP с липидными капельками в клетках листа.
Пример 12. Сайленсинг генов TGD в растениях
Согласно оценкам Li-Beisson et al. (2013), в листьях Arabidopsis (растения 16:3) приблизительно 40% жирных кислот, синтезированных в хлоропластах, поступают в прокариотический путь, тогда как 60% экспортируются для поступления в эукариотический путь. После того десатурации в ER, приблизительно половины указанных экспортированных жирных кислот возвращается в пластиду, чтобы обеспечить синтез галактолипидов для тилакоидных мембран. В транспорте (импорт) жирных кислот в пластиду, в виде DAG или фосфолипидов, принимает участие TGD1, пермеаза-подобный белок внутренней оболочки хлоропласта. Транспортер липидов ABC Arabidopsis, содержащий белки TGD1, 2 и 3, идентифицирован Benning et al. (2008 и 2009), и позже Roston et al. (2012). Указанный комплекс белка локализован во внутренней мембране оболочки хлоропласта, и предполагается, что он опосредует перенос фосфатидата через указанную мембрану. Полипептид TGD2 представляет собой фосфатидил-связывающий белок, и TGD3 представляет собой АТФазу. Новый белок Arabidopsis, TGD4, идентифицирован с помощью генетического подхода (Xu et al., 2008), где инактивация гена TGD4 также блокирует перенос липида из ER в пластиды. Последние биохимические данные указывают на то, что TGD4 является фосфатидат-связывающим белком, расположенным во внешней мембране оболочки хлоропласта (Wang и Benning, 2012).
Xu et al. (2005) описали подтекающие аллели tgd1 в A. thaliana, приводящие к снижению роста растения и высокой частоте абортов эмбриона. Листовая ткань мутантов tgd1 A. thaliana содержит повышенные уровни TAG, вероятно в форме цитозольных масляных капелек. Кроме того, повышенные уровни TAG обнаружены в корнях мутантов tgd1. Не было обнаружено отличий в содержании масла в семенах. О подобной аккумуляции TAG в ткани листа сообщалось для мутантов A. thaliana tgd2 (Awai et al., 2006), tgd3 (Lu et al., 2007) и tgd4 (Xu et al., 2008). Все мутантные аллели tgd были достаточно неплотными или значительно ослабляли развитие растения.
Сайленсинг TGD1
Конструкция сайленсинга, направленная против пластидного импортера TGD1, сгенерирована на основании полноразмерного транскрипта мРНК, идентифицированного в транскриптоме N. benthamiana. Фрагмент размером 685 пар оснований амплифицируют из кДНК листа N. benthamiana при инкорпорации в сайт PmlI на 5' конце. Сначала фрагмент TGD1 клонируют в pENTR/D-TOPO (Invitrogen), и затем вставляют в вектор назначения pHELLSGATE12 посредством клонирования LR (Gateway). Полученный вектор экспрессии, обозначенный как pOIL025, временно экспрессируют в N. benthamiana, чтобы оценить влияние сайленсинга гена TGD1 на уровни TAG в листьях. Конструкцию шпильки TGD1 размещают под контролем индуцибельного промотора alcA A. niger посредством субклонирования в виде фрагмента Pml I-EcoRV в сайты NheI (Кленова)-SfoI pOIL020 (ниже). Полученным вектором, обозначенным как pOIL026, супертрансформируют гомозиготную линию N. tabacum pJP3502 с целью дополнительного повышения уровней масла в листьях.
Получены дополнительные конструкции для экспрессии шпилечной РНК с целью снижения экспрессии генов TGD-2, -3, -4 и -5. С применением указанных конструкций получены трансформированные растения, и в трансформантах определено содержание масла. Трансформированные растения скрещены с трансформантами, полученными с использованием pJP3502 или других комбинаций генов, как описано выше.
Пример 13. Экспрессия комбинаций генов в картофельных клубнях
Конструкция pJP3506
Генетическая конструкция, содержащая три гена для экспрессии в картофельных клубнях, получена и применим для трансформации картофеля. Указанная конструкция обозначена как pJP3506 и базируется на существующем векторе pJP3502 (WO2013/096993) с заменой промоторов, чтобы обеспечить специфическую для клубня экспрессию. pJP3506 содержит (i) ген устойчивости к канамицину NPTII под управлением промотора 35S с удвоенным участком энхансера (e35S), в качестве гена селективного маркера, и три экспрессионные кассеты генов, которые представляют собой (ii) 35S::AtDGAT1, кодирующую DGAT1 Arabidopsis thaliana, (iii) B33::AtWRI1, кодирующую WRI1 Arabidopsis thaliana, и (iv) B33::олеозин кунжута, кодирующую олеозин из Sesame indicum. Нуклеотидные последовательности, кодирующие указанные полипептиды, являются такими же, как в pJP3502. Промотор пататина B33 (B33) представляет собой специфический для клубня промотор, полученный из Solanum tuberosum, который был предложен д-ром Alisdair Fernie, Институт молекулярной физиологии растений им. Макса Планка, Потсдам, Германия. Круговая карта плазмиды pJP3506 представлена на Фигура 17.
Последовательность промотора пататина B33 S. tuberosum, применяемая в конструкции pJP3506, представляет собой укороченную версию, в которой 183 нуклеотида удалены на 5' конце и 261 нуклеотид удален на 3' конце, по сравнению с номером доступа GenBank X14483. Нуклеотидная последовательность промотора пататина B33, применяемого в pJP3506, приведена в SEQ ID NO: 202.
Трансформация картофеля
Саженцы картофеля (Solanum tuberosum), сорт Atlantic, выращенные в культуре ткани, в асептических условиях, приобретены у Toolangi Elite, сертифицированного органа семенного картофеля в Виктории (СОСКВА), Австралия. Междоузлия стебля разрезают на куски длиной приблизительно 1 см под суспензией штамма LBA4404 Agrobacterium tumefaciens, содержащего pJP3506. Клетки Agrobacterium выращивают до OD 0,2 и разбавляют равным объемом среды МС. Избыток суспензии Agrobacterium удаляют кратковременным промоканием куском стебля стерильной фильтровальной бумагой, с последующим переносом на среду МС и инкубированием при 24°C в течение двух суток (сокультивирование). Затем междоузлия переносят на свежую среду МС с добавлением 200 мкг/л НУК, 2 мг/л бензиламинопурина (BAP) и 250 мг/л цефотаксима. Селекцию трансгенных каллюсов начинают через 10 суток, после переноса междоузлий на свежую среду МС с добавлением 2 мг/л BAP, 5 мг/л GA3, 50 мг/л канамицина и 250 мг/л цефотаксима. Побеги, регенерирующие из каллюсов, вырезают и помещают на простую среду МС для индукции корней, перед пересадкой в горшок диаметра 15 см, содержащий почвенную смесь и выращиванием в теплице до состояния зрелости растения, включая рост клубня.
Экстракция ДНК и молекулярная идентификация трансгенных растений способом ПЦР
Диски диаметром приблизительно 1 см получают из листьев картофеля от растений в теплице. Их помещают в планшет для микротитрования с глубокими лунками и лиофилизируют в течение 48 часов. Далее лиофилизированные образцы листьев измельчают в порошок, добавляя стальной шарик в каждую лунку, и встряхивают планшет в лизаторе для тканей Reicht (Qiagen) с максимальной частотой 28/с в течение 2 минут для каждой стороны планшета для микротитрования. 375 мкл экстракционного буфера, содержащего 0,1 М Трис-HCl, pH 8,0, 0,05 М ЭДТА и 1,25% SDS, добавляют в каждую лунку, содержащую порошкообразную листовую ткань. Через 1 час инкубации при 65°C, 187 мкл 6 М ацетата аммония добавляют в каждую лунку, и смеси выдерживают при 4°C в течение 30 минут перед центрифугированием планшетов на протяжении 30 минут при 3000 об/мин. 340 мкл супернатанта из каждой лунки переносят в новый планшет для микротитрования с глубокими лунками, содержащий 220 мкл изопропанола и выдерживают в течение 5 минут при комнатной температуре перед центрифугированием при 3000 об/мин на протяжении 30 минут. Осажденные гранулы ДНК промывают 70% этанолом, сушат на воздухе и ресуспендируют в 225 мкл H2O на образец.
По два мкл каждого препарата ДНК образцов листьев добавляют к 20 мкл реакционной смеси ПЦР с помощью системы ПЦР HotStar (Qiagen). Пару олигонуклеотидных праймеров, основанных на 5' и 3' последовательностях из гена WRI1 Arabidopsis thaliana, кодон-оптимизированного для табака, применяют в реакциях ПЦР. Последовательности праймеров: Nt-Wri-P3: 5'-CACTCGTGCTTTCCATCATC-3' (SEQ ID NO: 203) и Nt-Wri-P1: 5'-GAAGGCTGAGCAACAAGAGG-3'(SEQ ID NO: 204). Кроме того, пару олигонуклеотидных праймеров, основанных на гене DGAT1 Arabidopsis thaliana, кодон-оптимизированном для табака, применяют в отдельной реакции ПЦР для каждого образца ДНК. Последовательности праймеров: Nt-DGAT-P2: 5'-GGCGATTTTGGATTCTGC-3' (SEQ ID NO: 205) и Nt-DGAT-P3: 5'-CCCAACCCTTCCGTATACAT-3' (SEQ ID NO: 206). Амплификацию проводят с начальным циклом при 95°C в течение 15 минут, за которым следуют 40 циклов при 95°C в течение 30 секунд, при 57° C в течение 30 с и при 72°C в течение 60 секунд. Продукты ПЦР обрабатывают электрофорезом на 1% агарозном геле для обнаружения специфических продуктов амплификации.
Анализ липида картофельных клубней
Тонкие ломтики клубней, собранные с регенерированных картофельных растений для подтвержденных трансгенных растений и нетрансформированного контроля, лиофилизируют в течение 72 часов, и анализируют содержание и состав липида. Общие липиды экстрагируют из сухих тканей клубня с использованием хлороформа: метанола: 0,1 М KCl (2:1:1 об/об/об), как указано ниже. Лиофилизированные ткани клубня сначала гомогенизируют в хлороформе: метаноле (2:1, об/об) в пробирке Эппендорфа, содержащей металлический шар, с помощью лизатора для тканей Reicht (Qiagen) в течение 3 минут при частоте 29/с. После смешивания каждого гомогената с Vibramax 10 (Heidolph) при 2000 об/мин в течение 15 минут, к каждому образцу добавляют 1/3 объема 0,1 М раствора KCl и дополнительно перемешивают. После центрифугирования при 10000 g в течение 5 минут, из каждого образца отбирают нижнюю фракцию, содержащую липиды, и полностью выпаривают в потоке N2. Каждый препарат липида растворяют в 3 мкл CHCl3 на миллиграмм сухой массы клубня. Аликвоты препаратов липида наносят на пластину для тонкослойной хроматографии (TLC) (20 см x 20 см, силикагель 60, Merck) и элюируют гексаном: диэтиловым эфиром: уксусной кислотой (70:30:1, об/об/об). Пластину TLC обрызгивают примулином и визуализируют в УФ-свете, чтобы проявить пятна липида. TAG и PC извлекают, соскабливая кремния диоксид с подходящих полос, и превращают в метиловые сложные эфиры жирных кислот (FAME), инкубируя материал в 1 н метанольной HCl (Supelco, Bellefonte, PA) при 80°C в течение 2 часов, вместе с известным количеством тригептадеканоина (Nu-Chek PREP, Inc. USA) в качестве внутреннего стандарта для количественного определения липида. FAME анализируют на приборе GC-ПИД (7890A GC, Agilent Technologies, Palo Alto, CA), оборудованном колонкой BPX70 длиной 30 м (внутренний диаметр 0,25 мм, толщина пленки 0,25 мм, SGE, Austin, USA), как было описано ранее (Petrie et al., 2012). Пики интегрируют с помощью программного обеспечения Agilent Technologies ChemStation (версия B.04.03).
Среди приблизительно 100 регенерированных индивидуальных трансгенных линий, анализ липидов, полученных из молодых картофельных клубней диаметром приблизительно 2 см обнаружил повышенные уровни общих липидов, TAG и фракций фосфолипидов в клубнях многих трансгенных растений, где наблюдается диапазон от отсутствия повышения до значительного повышения. Первый анализ липидов картофельного клубня показал, что типичный картофельный клубень дикого типа в ранней стадии развития (диаметр приблизительно 2 см) содержит приблизительно 0,03% TAG, в пересчете на сухую массу.
Содержание общих липидов было повышено до 0,5-4,7% масс. (сухая масса) в клубнях 21 индивидуального трансгенного растения, представляющего 16 независимо трансформированных линий (Таблица 29). Клубни линии №69 продемонстрировали наиболее высокую аккумуляцию TAG, в среднем 3,3%, в пересчете на сухую массу. Это соответствует увеличению приблизительно в 100 раз относительно клубней дикого типа в такой же стадии развития. Дополнительно, клубни этой же трансгенной линии аккумулировали наиболее высокие из наблюдаемые уровней фосфолипидов, 1,0% масс. в молодых клубнях, в пересчете на сухую массу (Таблица 30). Кроме того, повышенная накопление липида сопровождалась модификацией состава жирных кислот в трансгенных клубнях. Трансгенные клубни систематически аккумулировали более высокие проценты насыщенных и мононенасыщенных жирных кислот (MUFA) и более низкие уровни полиненасыщенных жирных кислот (PUFA) в общем содержании жирных кислот и во фракции TAG общих жирных кислот (Таблица 29), конкретно, сниженный уровень 18:3 (ALA), который был уменьшен с приблизительно 17% для дикого типа до менее чем 10% в трансгенных клубнях. Уровень олеиновой кислоты (18:1) в общем содержании жирных кислот повышался с приблизительно 1% для дикого типа до более чем 5% во многих линиях и более чем 15% в некоторых клубнях. Хотя уровни пальмитиновой кислоты были повышены, уровни стеариновой кислоты (18:0) были сниженные в лучших трансгенных линиях (Таблица 28 и 29).
Таблица 28. Общий выход липида (% масс. от сухой массы картофельного клубня) и жирнокислотный состав в характерных молодых клубнях картофеля, трансформированных T-ДНК pJP3506, до цветения растений. Клубни линии 65 были эквивалентны клубням дикого типа (нетрансгенным).
d11
Таблица 29. Выход TAG (% масс. от сухой массы картофельного клубня) и его жирнокислотный состав в характерных молодых клубнях картофеля, трансформированных T-ДНК pJP3506, до цветения растений.
d11
Таблица 30. Выход фосфолипидов (% масс. от сухой массы картофельного клубня) и их жирнокислотный состав в характерных молодых клубнях картофеля, трансформированных T-ДНК pJP3506, до цветения.
d11
Трансгенные растения картофеля поддерживали в теплице, чтобы обеспечить непрерывный рост клубней. Более крупные клубни линии №69 имели более высокие уровни TFA и TAG, чем клубни диаметром приблизительно 2 см.
Дополнительно увеличенные уровни TFA и TAG получены в клубнях картофеля посредством добавления химерного гена, который кодирует осуществляющую сайленсинг РНК для понижающей регуляции экспрессии эндогенного гена SDP1, в комбинации с генами WRI1 и DGAT.
Дополнительные комбинации генов для трансформации картофеля
Тотальную РНК из свежих развивающихся клубней картофеля (Solanum tuberosum L. cv. Atlantic) экстрагировали способом с TRIzol (Invitrogen). Избранные области кДНК, кодирующих малую субъединицу АГФазы картофеля и SDP1, получали посредством ОТ-ПЦР с использованием следующих праймеров: st-AGPs1: 5'-ACAGACATGTCTAGACCCAGATG-3' (SEQ ID NO: 242), st-AGPa1: 5'-CACTCTCATCCCAAGTGAAGTTGC-3' (SEQ ID NO: 243); st-SDP1-s1: 5'-CTGAGATGGAAGTGAAGCACAGATG-3' (SEQ ID NO: 244), и st-SDP1-a1: 5'-CCATTGTTAGTCCTTTCAGTC-3' (SEQ ID NO: 245). Затем продукты ПЦР очищали и лигировали в pGEMT Easy.
После верификации секвенированием ДНК, клонированные продукты ПЦР непосредственно применяют в качестве последовательности гена-мишени, с целью получения конструкции шпилечной РНКи, или сливают посредством ПЦР с перекрывающимися праймерами. Три фрагмента ПЦР (SDP1, АГФаза, SDP+АГФ) в дальнейшем клонируют в вектор pKannibal, который содержит специфические рестрикционные сайты, для клонирования желательного фрагмента в смысловой и антисмысловой ориентации. Выбранные рестрикционные сайты представляют собой BamHI и HindIII для клонирования фрагмента в смысловой ориентации и KpnI и XhoI для вставки фрагмента в антисмысловой ориентации. Наборы праймеров, применяемые для амплификации трех фрагментов гена-мишени, модифицируют добавлением рестрикционных сайтов, которые направляют фрагмент в клонирующие сайты pKannibal. Экспрессионные кассеты, содержащие фрагмент-мишень ДНК между промотором 35S и терминатором OCS в pKannibal, высвобождают с помощью Not1 и клонируют в бинарный вектор pWBVec2, с гигромицином в качестве селективного маркера для растений. Такие бинарные векторы вводят в штамм AGL1 A. tumefaciens и применяют для трансформации картофеля, как описано выше.
Пример 19. Модификация признаков в однодольном растении
Экспрессия в эндосперме
Содержание масла в эндосперме однодольных видов растений Triticum aestivum (пшеница) и, таким образом, в зерне растений, повышают экспрессию комбинации генов, кодирующих WRI1, DGAT и олеозин, в эндосперме в ходе развития семени с использованием специфических для эндоспермы промоторов. Конструкция (обозначенная как pOIL-Endo2) содержит химерные гены: (a) промотор гена Glu1 Brachypodium distachyon::кодирующий белок участок гена Zea mays, кодирующего полипептид ZmWRI1 (SEQ ID NO: 35)::участок терминатора/полиаденилирования из гена лектина Glycine max, (b) промотор гена глютенина Bx17 Triticum aestivum::кодирующий белок участок гена A. thaliana, кодирующего полипептид AtDGAT1 (SEQ ID NO: 1)::участок терминатора/полиаденилирования из гена Nos Agrobacterium tumefaciens, (c) промотор гена GluB4 Oryza sativa::кодирующий белок участок гена Sesame indicum, кодирующего участок полипептида олеозина::участок терминатора/полиаденилирования из гена лектина Glycine max и (d) промотор 35S::участок, кодирующий устойчивость к гигромицину, в качестве гена селективного маркера. Конструкцию применяют для трансформации незрелых эмбрионов T. aestivum (сорт Fielder) посредством опосредованной Agrobacterium трансформации. Инокулированные незрелые эмбрионы подвергают воздействию гигромицина, чтобы отобрать трансформированные побеги, а затем переносят в среду корнеобразования для формирования корней, перед переносом в грунт.
Получены тридцать трансформированных растений, на которых завязалось семя T1 и которые содержат T-ДНК из pOIL-Endo2. Урожай зрелых семян собран со всех 30 растений, и 6 семян каждого семейства разрезаны пополам. Половинки, содержащие эмбрион, хранили для последующего проращивания; другую половинку, содержащую по большей части эндосперм, экстрагировали и оценивали содержание масла. T-ДНК, вставленная в геном пшеницы, все еще сегрегируется в семенах T1 указанных растений, поэтому семена T1 представляют собой смесь гомозиготных трансформированных, гетерозиготных трансформированных и нулевых по T-ДНК. Повышенное содержание масла наблюдается в эндосперме некоторых из семян, где некоторые семена демонстрируют увеличение уровней TAG более, чем в 5 раз. В половинках с эндоспермом шести семян (сорт Fielder) дикого типа содержание TAG составляет приблизительно 0,47% масс. (диапазон от 0,37% до 0,60%), по сравнению с содержанием TAG 2,5% в некоторых зернах. В некоторых семействах все шесть семян содержат свыше 1,7% TAG; другие очевидно сегрегированы между диким типом и повышенным содержанием TAG. Кроме того, в эндосперме с повышенным содержанием TAG состав жирных кислот модифицирован, демонстрируя увеличение процентного содержания олеиновой кислоты и пальмитиновой кислоты и снижение процентного содержания линолевой кислоты (Таблица 31). Семя T1 беспрепятственно прорастало с той же скоростью, что и семя соответствующего дикого типа, где растения, представляющие индивидуумы как с высоким содержанием масла, так и с низким содержанием масла из 14 семейств T0 выращены до состояния зрелости. Указанные растения имели полноценную мужскую и женскую фертильность.
Таблица 31. Состав жирных кислот (% от общих жирных кислот) в TAG и общее содержание TAG (% масс. масла в половинке с эндоспермом) в трансгенном эндосперме пшеницы
Анализировали 220 семян T2 из 22 отобранных растений T1, плюс 40 растений из 3 различных родительских растений Fielder. В большинстве случаев, тестировали десять семян T2 из каждого растения T1. Некоторые из отобранных растений T1 являлись нулевыми с соответствующими дикому типу уровнями TAG в эндосперме. Некоторые из результатов анализа половинок семян с эндоспермом представлены на Фигуре 18. Растения T1 с высоким содержанием масла в эндосперме образовывали семена T2, многие из которых имели увеличенное содержание масла в эндосперме, в то время как контрольные растения Fielder и являющиеся нуль-сегрегантами растения T1 образовывали семена со сходными уровнями масла в эндосперме (общим содержанием жирных кислот, TFA).
Зерно можно использовать для получения пищевых продутов для употребеления человеком или в качестве корма для животных, обеспечивая зерно с увеличенным содержанием энергии на единицу массы (плотностью энергии) и получая в результате увеличенные темпы прироста у животных например, таких как домашняя птица, свиньи, крупный рогатый скот, овцы и лошади.
Дополнительно, конструкцию pOIL-Endo2 применяют для трансформации кукурузы (Zea mays) и риса (Oryza sativa), с целью получения трансгенных растений с повышенным содержанием TAG в эндосперме и, таким образом, в зерне.
Экспрессия в листьях и стеблях
Серия бинарных векторов экспрессии разработана для опосредованной Agrobacterium трансформации сорго (S. bicolor) и пшеницы (Triticum aestivum), с целью повышения содержания масла в вегетативных тканях. Исходными векторами для конструкций являются pOIL093-095, pOIL134 и pOIL100-104 (см. Пример 5). Сначала, фрагмент ДНК, кодирующий опосредованной WRI1 Z. mays, амплифицируют способом ПЦР с использованием pOIL104 в качестве шаблона и праймеров, содержащих рестрикционные сайты KpnI. Указанный фрагмент субклонируют в направлении по ходу транскрипции относительно конститутивного промотора Actin1 Oryza sativa в pOIL095, используя сайт KpnI. Результирующий вектор обозначен как pOIL154. Фрагмент ДНК, кодирующий DGAT2a Umbelopsis ramanniana под контролем промотора убиквитина Z. mays (pZmUbi), выделяют из pOIL134 в виде фрагмента NotI и вставляют в сайт NotI pOIL154, с получением pOIL155. Экспрессионную кассету, состоящую из кодирующего PAT участка под контролем промотора pZmUbi, фланкированного на 3' конце участком терминатора/полиаденилирования NOS A. tumefaciens, конструируют амплификацией кодирующего PAT участка с использованием pJP3416 в качестве шаблона. Разработаны праймеры для инкорпорации рестрикционных сайтов BamHI и SacI на 5' и 3' конце, соответственно. После двойного расщепления BamHI+SacI, фрагмент PAT клонируют в соответствующие сайты pZLUbi1casNK. Полученное промежуточное соединение обозначено как pOIL141. Далее, кассету селективного маркера PAT вводят в остов pOIL155. В этой точке pOIL141 сначала разрезают с помощью NotI, фрагмента Кленова ДНК полимеразы I с тупыми концами, и далее расщепляют с помощью AscI. Затем полученный фрагмент размером 2622 пар оснований субклонируют в сайты ZraI-AscI pOIL155, с получением pOIL156. В конце, промотор Actin1, управляющий экспрессией WRI1 pOIL156, обменивают на промотор маленькой субъединицы Rubisco Z. mays (pZmSSU), с получением pOIL157. Данный вектор получен ПЦР амплификацией промотора SSU Z. mays, с использованием pOIL104 в качестве шаблона и фланкирующих праймеров, содержащих рестрикционные сайты Asi SI и PmlI. Далее полученный ампликон разрезают с помощью SpeI+MluI и субклонируют в соответствующие сайты pOIL156.
Указанные векторы таким образом содержат следующие кассеты экспрессии:
pOIL156: промотор актина 1 O. sativa::WRI1 Z. mays, промотор убиквитина Z. mays::DGAT2a U. rammaniana и промотор убиквитина Z. mays::PAT
pOIL157: промотор SSU Z. mays::WRI1 Z. mays, промотор убиквитина Z. mays::DGAT2a U. rammaniana и убиквитина Z. mays::PAT.
Вторые серии бинарных векторов экспрессии, содержащих промотор старения SEE1 Z. mays (Robson et al., 2004, см. Пример 5), фактор транскрипции LEC1 Z. mays (Shen et al., 2010) и фрагмент SDP1 шпРНКи S. bicolor, сконструированы, как указано ниже. Сначала участок прикрепления к матриксу (MAR) вводят в pORE04 посредством расщепления pDCOT с помощью AatII+ SnaBI, и субклонирования в сайты AatII+ EcoRV pORE04. Полученный промежуточный вектор обозначен как pOIL158. Далее ген селективного маркера PAT под контролем промотора убиквитина Z. mays субклонируют в pOIL158. К этому времени, pOIL141 сначала расщеплен NotI, обработан фрагментом Кленова ДНК полимеразы I и в конце расщеплен AscI. Полученный фрагмент вставляют в сайты AscI+ ZraI pOIL158, с получением pOIL159. Оригинальный источник репликации RK2 oriV в pOIL159 заменяют источником RiA4 посредством рестрикционного расщепления pJP3416 с помощью SwaI+ SpeI, с последующим субклонированием в сайты SwaI+AvrII pOIL159. Полученный вектор обозначен как pOIL160. Синтезирован фрагмент «part1 старения однодольных» размером 10019 тыс. пар оснований, содержащий следующие кассеты экспрессии: актин 1 O. sativa::DGAT1 A. thaliana, кодон-оптимизированный для экспрессии в Z. mays, SEE1Z. mays::WRI1Z. mays, SEE1 Z. mays::LEC1Z. mays. Данный фрагмент субклонируют в виде фрагмента SpeI-EcoRV в сайты SpeI-StuI pOIL160, с получением pOIL161. Синтезирован второй фрагмент « part2 старения однодольных» размером 7967 тыс. пар оснований, который содержит следующие элементы: MAR, убиквитин Z. mays::шпРНКи, фрагмент, нацеленный на S. bicolor/SDP1 T. aestivum, пустая кассета под контролем промотора актина 1 O. sativa. Последовательности двух TAG-липаз SDP1 S. bicolor (номера доступа XM_002463620; SEQ ID NO. 233 и XM_002458486; SEQ ID NO: 169) и одна последовательность SDP1 T. aestivum (номер доступа AK334547) (SEQ ID NO: 234) получены способом поиска BLAST с последовательностью SDP1 A. thaliana (номер доступа NM_120486). Разработана конструкция синтетической шпильки (SEQ ID NO: 235), содержащая четыре фрагмента (67 пар оснований, 90 пар оснований, 50 пар оснований, 59 пар оснований) последовательности XM_002458486 S. bicolor, которая продемонстрировала наиболее высокую идентичность с последовательностью SDP1 T. aestivum. Кроме того, включен фрагмент размером 278 пар оснований, полученный из липазы SDP1 S. bicolor XM_002463620, с целью усиления эффективности сайленсинга в отношении обеих последовательностей SDP1 S. bicolor. Фрагмент «part2 старения однодольных» субклонируют в виде фрагмента BsiWI-EcoRV в сайты BsiWI-FspI pOIL161. Полученный вектор обозначен pOIL162.
Генетические конструкции pOIL156, pOIL157, pOIL161 и pOIL162 применяют для трансформации S. bicolor и T. aestivum с использованием опосредованной Agrobacterium трансформации. Трансгенные растения отобраны на основе устойчивости к гигромицину и содержат повышенные уровни TAG и TFA в вегетативных тканях, по сравнению с нетрансформированными контрольными растениями. Такие растения пригодны для питания животных в виде сена или силоса, а также можно использовать для получения зерна или экстракции масла.
Дополнительные генетические конструкции получены для экспрессии комбинаций полипептидов в листьях и стеблях однодольных растений, включая C4-фотосинтезирующие растения S. bicolor и Z. mays. Получено несколько конструкций, содержащих гены для экспрессии WRI1, DGAT и олеозина, с каждым геном под контролем конститутивного промотора, такого как промотор гена убиквитина маиса или промотор гена актина риса, и содержащих ген NPTII в качестве гена селективного маркера. В одной конкретной конструкции, WRI1 представляет собой WRI1 сорго. В другой, олеозин представляет собой олеозин L Si (см. Пример 17). В других конкретных конструкциях, ген олеозина заменен на ген, кодирующий либо LDAP2, либо LDAP3 из S. sebiferum (Пример 11). Эти конструкции используют в качестве «коровых конструкций» для трансформации S. bicolor и Z. mays и используют отдельно или в комбинации с генетическими конструкциями для экспрессии шпилечной РНК, нацеленной на один или несколько генов SDP1 в сорго или маисе (см. выше), с конструкцией, кодирующ Lec2 под контролем промотора SEE1 (специфичного для старения), или с обеими. Получена другая конструкция, содержащая три гена, а именно, для экспрессии шпилечной РНК, нацеленной на эндогенный ген TGD5 для уменьшения его экспрессии, тиоэстеразу жирного ацила FatA и PDAT, которую используют для увеличения уровня TAG и/или параметра TTQ для растений, трансформированных этой конструкцией.
Пример 15. Экстракция масла
Экстракция липида из листьев
Урожай трансгенных листьев табака, трансформированных T-ДНК из pJP3502, собирают с растений, выращенных в теплице на протяжении летних месяцев. Листья сушат, а затем измельчают до фрагментов размером 1-3 мм перед экстракцией. Измельченный материал обрабатывают экстракцией в аппарате Сокслета (кипячение с обратным холодильником) в течение 24 часов с выбранными растворителями, как описано ниже. 5 г материала сухих листьев табака и 250 мл растворителя применяют в каждом эксперименте с экстракцией.
Экстракция гексаном в качестве растворителя
Гексан обычно находит коммерческое применение в качестве растворителя для экстракции масла из прессованных семян масличных культур, например, рапса, где гексан экстрагирует нейтральные (неполярные) липиды, и в другой роли он применим впервые. Масса экстрагированного липида составила 1,47 г из 5 г листового материала, выход липида составил 29% масс. Проведен анализ экстрагированного гексаном липида в ДМСО способом 1H ЯМР. Анализ продемонстрировал сигналы, типичные для длинноцепочечных жирных кислот в триглицериде, без ароматических продуктов. Далее липид обрабатывают GCМС с целью идентификации основных компонентов. Анализ экстрагированного гексаном липида способом прямой GCМС провести сложно, поскольку температура кипения слишком высокая, и материал разлагается в ходе GCМС. В таких ситуациях общая техника анализа включает первоначальное получение метиловых сложных эфиров жирных кислот, которые были получены, как указано ниже: 18 мг липидного экстракта растворяют в 1 мл толуола, добавляют 3 мл сухого 3 н метанольного раствора HCL и перемешивают на протяжении ночи при 60°C. К охлажденному флакону добавляют 5 мл 5% NaCl и 5 мл гексана и встряхивают. Органический слой извлекают, и экстракцию повторяют еще с 5 мл гексана. Объединенные органические фракции нейтрализуют 8 мл 2% KHCO3, отделяют и сушат с помощью Na2SO4. Растворитель выпаривают в потоке N2, после чего концентрацию доводят до 1 мг/мл в гексане для анализа GCМС. Основными жирными кислотами являются 16:0 (пальмитиновая, 38,9%) и 18:1 (олеиновая, 31,3%).
Экстракция ацетоном в качестве растворителя
Ацетон применяют в качестве растворителя для экстракции, поскольку его свойства растворителя должны обеспечивать экстракцию практически всего липида из листьев, т.е. как неполярных, так и полярных липидов. Экстрагированное ацетоном масло выглядит подобным экстрагированному гексаном липиду. Масса экстрагированного липида составила 1,59 г из 5 г листьев табака, т.е. 31,8% масс. Проведен анализ липида в ДМСО способом 1H ЯМР. Наблюдаются сигналы, типичные для длинноцепочечных жирных кислот в триглицериде, сигналов ароматических продуктов.
Экстракция горячей водой в качестве растворителя
Осуществлена попытка экстракции горячей водой в качестве растворителя для экстракции, с целью определения ее пригодности для получения масла из листьев табака. Экстрагированный водой материал выглядит гелеподобным и желируется при охлаждении. Масса экстракта составила 1,9 г или 38% масс. Полученный материал подобен густому гелю и, вероятно, содержит полярные соединения из листьев, такие как сахара и другие углеводы. Проведен анализ материала в ДМСО способом 1H ЯМР. Анализ продемонстрировал сигналы, типичные для длинноцепочечных жирных кислот в триглицериде, и не показал экстрагированных ароматических продуктов. Лишний твердый материал был экстрагирован гексаном, давая 20% масс. липида, что указывает на неэффективность экстракции неполярных липидов водой.
Экстракция этанолом в качестве растворителя
Этанол применяли в качестве растворителя для экстракции, с целью определения его пригодности для получения масла из листьев табака. Экстрагированный этанолом липид выглядит подобным как липиду, экстрагированному водой, так и липиду, экстрагированному гексаном, он имеет желто-красный цвет, гелеподобный внешний вид и желируется при охлаждении. Масса экстрагированного липида составила 1,88 г из 5 г табака, или 37,6% масс. Кроме того, этанол в качестве растворителя экстрагировал из листьев табака некоторые полярные соединения.
Экстракция эфиром в качестве растворителя
Осуществлена попытка экстракции диэтиловым эфиром в качестве растворителя для экстракции, поскольку предполагалось, что он может экстрагировать меньшее количество примесей, чем другие растворители. В результате экстракции получено 1,4 г или 28% масс. Экстрагированный эфиром липид подобен материалу, экстрагированному гексаном, имеет желтоватый цвет и выглядит немного чище, чем гексановый экстракт. Несмотря на то, что полученный с диэтиловым эфиром экстракт выглядит наиболее чистым маслом, анализ способом ЯМР продемонстрировал смесь большего количества органических соединений.
Пример 16. Рацион кормления молочных коров
Листья и стебли из растений сорго или кукурузы, имеющие увеличенное содержание TAG и TFA, собирали и нарезали на фрагменты размером 1-2 см. Переработанные части растений силосовали в течение по меньшей мере двух недель и затем смешивали с другими компонентами для получения пищевого продукта для молочных коров. Питательная смесь для молочных коров содержит: 7,5-10 кг силоса сорго или кукурузы, содержащего увеличенное количество TAG и TFA, 4-5 кг люцернового сена, 1 кг пивной дробины (приблизительно 67% перевариваемого сухого вещества), 1-2 кг жмыховой муки (из канолы или сои) или хлопчатника, 0,5 кг мелассы и минеральные добавки, такие как кальций, фосфор, магний и сера. Липид оптимально присутствует на уровне 5-7% от общей массы сухого вещества. Можно добавлять дополнительные аминокислоты, такие как лизин и метионин, или небелковые источники азота, такие как мочевина, в зависимости от общего содержания белка. Пищевой продукт имеет увеличенную плотность энергии, увеличенную пищевую ценность, увеличенную питательную ценность и увеличенную перевариваемость по сравнению с соответствующим пищевым продуктом, полученным с использованием эквивалентного количества силоса из сорго или кукурузы дикого типа. Увеличенное содержание липидов в силосе из сорго или кукурузы с высоким содержанием масла приводит к дополнительной продукции молока вплоть до 3 литров в сутки и к увеличению на 0,33% жирности молока на каждый килограмм съеденного липида.
Телка может съедать эквивалент приблизительно 2,3% массы ее тела ежесуточно, и взрослая недойная корова может съедать эквивалент приблизительно 1,5% массы ее тела ежесуточно. Например, телка массой 300 кг может съедать вплоть до 7 кг сухого вещества, и взрослая недойная корова массой 470 кг может съедать приблизительно такое же количество. Дойные коровы имеют более высокое потребление корма, вплоть до приблизительно 4% массы тела в сутки. Действительно, потребление корма в пересчете на массу имеет тенденцию к увеличению с увеличением качества и вкусовой привлекательности корма.
Пример 17. Экспрессия белков масляных телец в вегетативных тканях растений
Кодирующую область белка, кодирующую ассоциированный с липидными капельками белок TadA Rhodococcus opacus (MacEachran et al. 2010; номер доступа HM625859), кодон-оптимизированную для экспрессии в двудольных растениях, таких как Nicotiana benthamiana, синтезировали в форме NotI-SpeI фрагмента ДНК. Фрагмент вставляли ниже промотора 35S в pJP3343 с использованием сайтов NotI-SpeI. Полученная плазмида обозначена pOIL380. Кодирующую область белка, кодирующую ассоциированный с липидными капельками белок олеозин L Sesame indicum (Tai et al. 2002; номер доступа AF091840; SEQ ID NO:305) синтезировали в форме NotI-SacI фрагмента ДНК и вставляли ниже промотора 35S в pJP3343 с использованием таких же сайтов. Полученная плазмида обозначена pOIL382. Кодирующую область белка, кодирующую ассоциированный с липидными капельками белок олеозин H1 Sesame indicum (Tai et al., 2002; номер доступа AF302807) синтезировали в форме NotI-SacI фрагмента ДНК и клонировали ниже промотора 35S в pJP3343 с использованием таких же сайтов. Полученная плазмида обозначена pOIL383. Вариант кодирующей белок области, кодирующий олеозин H1 S. indicum, имеющий три замены аминокислот для удаления сайтов убиквитинилирования (K130R, K143R, K145R) (Hsiao and Tzen, 2011) получали посредством направленного мутагенеза. Кодирующую область вставляли ниже промотора 35S в pJP3343 в форме фрагмента NotI-SacI. Полученная плазмида обозначена pOIL384. Кодирующую область белка, кодирующую ассоциированный с липидными капельками белок олеозин U1 листьев Vanilla planifolia (Huang and Huang, 2016; номер доступа SRX648194), кодон-оптимизировали для экспрессии в N. benthamiana, синтезировали в форме SpeI-EcoRI фрагмента ДНК и вставляли ниже промотора 35S в pJP3343 с использованием таких же сайтов. Полученная плазмида обозначена pOIL386. Кодирующую область белка, кодирующую ассоциированный с липидными капельками белок олеозин M мезокарпия Persea americana (Huang and Huang 2016; номер доступа SRX627420), кодон-оптимизировали для экспрессии в N. benthamiana, синтезировали в форме SpeI-EcoRI фрагмента ДНК и вставляли ниже промотора 35S промотор в pJP3343 с использованием таких же сайтов рестрикции. Полученная плазмида обозначена pOIL387. Кодирующую область белка, кодирующую ассоциированный с липидными капельками белок олеозин 3 Arachis hypogaea (Parthibane et al., 2012a; номер доступа AY722696), кодон-оптимизировали для экспрессии в N. benthamiana, фланкировали сайтами NotI и вставляли в бинарный экспрессирующий вектор pJP3502. Полученную плазмиду, pOIL041, расщепляли с использованием NotI, и полученный фрагмент ДНК 520 пар оснований вставляли ниже промотора 35S pJP3343. Полученная плазмида обозначена pOIL190. Подобным образом, кодирующую область для ассоциированного с липидными капельками белка калеозина 3 A. thaliana (Shen et al., 2014; Laibach et al., 2015; номер доступа AK317039) кодон-оптимизировали для экспрессии в N. benthamiana, фланкировали сайтами NotI и вставляли в pJP3502. Полученную плазмиду, pOIL042, расщепляли с использованием NotI, и полученный фрагмент ДНК 604 пар оснований вставляли ниже промотора 35S в pJP3343. Полученная плазмида обозначена pOIL191. Кодирующую область белка, кодирующую ассоциированный с липидными капельками белок стеролеозин A. thaliana (номер доступа AT081653) кодон-оптимизировали для экспрессии в N. benthamiana, фланкировали сайтами NotI и вставляли в pJP3502. Полученную плазмиду, pOIL043, расщепляли с использованием NotI, и полученный фрагмент ДНК 1069 пар оснований вставляли ниже промотора 35S в pJP3343. Полученная плазмида обозначена pOIL192. Кодирующую область белка, кодирующую белок масляных телец LSDP Nannochloropsis oceanica (Vieler et al., 2012; номер доступа JQ268559) кодон-оптимизировали для экспрессии в N. benthamiana, фланкировали сайтами NotI и вставляли в бинарный экспрессирующий вектор pJP3502. Полученную плазмиду, pOIL044, расщепляли с использованием NotI, и фрагмент ДНК 496 пар оснований вставляли ниже промотора 35S в pJP3343. Полученная плазмида обозначена pOIL193. Кодирующую область белка, кодирующую гидрофобин HFBI Trichoderma reesei (Linder et al., 2005; номер доступа Z68124) кодон-оптимизировали для экспрессии в N. benthamiana, фланкировали сайтами NotI и вставляли в pJP3502. Полученную плазмиду, pOIL045, расщепляли с использованием NotI и фрагмент ДНК 313 пар оснований вставляли ниже промотора 35S в pJP3343. Полученная плазмида обозначена pOIL194. Нацеленный на ER вариант гидрофобина HFBI Trichoderma reesei получали посредством изменения до пептида KDEL для удержания в ER на C-конце (Gutierrew et al., 2013). Этот вариант кодон-оптимизировали для экспрессии в N. benthamiana и клонировали в форме NotI фрагмента в pJP3502, получая pOIL046. Затем, pOIL046 расщепляли с использованием NotI, и фрагмент 325 пар оснований вставляли в pJP3343. Полученный вектор обозначен pOIL195.
Каждую из генетических конструкций, кодирующих ассоциированный с липидными капельками полипептиды, вводили в листья N. benthamiana в комбинации с генетическими конструкциями, кодирующими WRI1, DGAT1 и p19, как описано в Примере 1, с некоторыми незначительными модификациями. Культуры Agrobacterium tumefaciens, содержащие ген, кодирующий белок-супрессор сайленсинга p19, и представляющие интерес химерные гены, смешивали таким образом, что конечная OD600 каждой культуры была равной 0,125 перед инфильтрацией. Сравниваемые образцы были локализованы на одном и том же листе. После инфильтрации, растения N. benthamiana выращивали в течение дополнительных пяти суток перед сбором листовых дисков, пулировали в пределах трех листьев с одного и того же растения, лиофилизировали, взвешивали и хранили при -80°C. Общие липиды экстрагировали из лиофилизированных тканей с использованием хлороформа:метанола:0,1 M KCl (2:1:1 об./об./об.), и аликвоты наносили на пластину для тонкослойной хроматографии (TLC) и проявляли в гексане:диэтиловом эфире:уксусной кислоте (70:30:1, об./об./ об.). TAG выделяли, переводили в FAME в присутствии известного количества тригептадеканоина (Nu-Chek PREP, Inc. USA) в качестве внутреннего стандарта для количественной оценки липидов, и анализировали посредством GC-FID.
Анализы показали диапазон уровней TAG по сравнению с контролем WRI1+DGAT1. Некоторые конструкции, кодирующие ассоциированные с липидными капельками полипептиды, увеличивали уровень TAG по сравнению с контролем в некоторых анализах, в то время как другие не увеличивали. Постоянное и статистически значимое увеличение содержания TAG наблюдали, когда вводили конструкцию, экспрессирующую олеозин L Si (pOIL382) (Фигура 20); эта конструкция превосходила все другие, тестированные в этих анализах. Увеличение уровней C18:2 и C18:1 и уменьшение C16:0 также наблюдали в TAG для этой конструкции, по сравнению с контролем p19+WRI1+DGAT1 (Фигура 20). Микроскопические анализы для визуализации липидных капелек в клетках листьев, экспрессирующих олеозин L Si, показали уменьшение размера липидных капелек и увеличение распространенности, по сравнению с контролем.
Проводили дополнительные анализы с использованием радиоактивно меченного [14C]-ацетата для измерения скорости синтеза TAG для различных комбинаций генов, включающих каждый из ассоциированных с липидными капельками полипептидов. Проводили инфильтрацию [14C]-ацетат в те же самые ткани листьев на 3 сутки после инфильтрации генетических конструкций, т.е. после экспрессии генов в течение трех суток. Через три часа, листовые диски собирали, и общие липиды из тканей экстрагировали и фракционировали посредством TLC. Уровень радиоактивности в различных типах липидов количественно оценивали с использованием с использованием устройства для визуализации фосфора Fujifilm FLA-5000. Эти анализы показали увеличение скорости синтеза TAG в листьях, экспрессирующих олеозин L Si (pOIL382), так же как увеличение скорости синтеза PC и PA в течение трех часов в листьях, экспрессирующих олеозин L Si. В отличие от этого, для генетических конструкций, кодирующих олеозин H Si, олеозины листьев ванили и мезокарпия авокадо, не показали значительного эффекта на скорость синтеза или содержание TAG.
Специалисту в данной области понятно, что могут быть осуществлены многочисленные варианты и/или модификации изобретения, как проиллюстрировано в конкретных вариантах осуществления, без отклонения от содержания или объема изобретения, как было подробно описано. Таким образом, представленные варианты осуществления следует во всех отношениях интерпретировать как иллюстративные, но не ограничивающие.
Полное содержание всех публикаций, обсуждаемых и/или цитируемых в настоящем описании, приведено в настоящем описании в качестве ссылки.
Любое обсуждение документов, действий, материалов, устройств, изделий или подобного, включенное в настоящий документ, служит исключительно цели обеспечения контекста для настоящего изобретения. Это не следует интерпретировать как допущение того, что любые или все из этих материалов образуют часть предшествующего уровня техники или были общедоступным знанием в данной области, относящимся к настоящему изобретению, поскольку они существовали до даты приоритета каждого из пунктов формулы изобретения в этой заявке.
ССЫЛКИ:
Alemanno et al. (2008) Planta 227:853-866.
Almeida and Allshire (2005) TRENDS Cell Biol. 15:251-258.
Alonso et al. (2009) Plant Cell 21: 1747-1761.
Alonso et al. (2010) Green Chem. 12:1493-1513.
Alvarez et al. (2000) Theor. Appl. Genet. 100:319-327.
Andre at al (2012) Proc. Natl. Acad. Sci. U.S.A. 109:10107-10112.
Andrianov et al. (2010) Plant Biotech. J. 8:277-287.
Awai et al (2006) Biochem. Soc. Trans. 34:395-398.
Bartlett et al. (2008) Plant Methods 4:22.
Bates (2016). Biochim et Biophys Acta 1961:1214-1225.
Bates and Browse (2011). Plant J. 68:387-399.
Baud et al. (2007) Plant J. 50:825-838.
Baud and Lepiniec (2010) Progr. Lipid Res. 49: 235-249.
Baumlein et al. (1991) Mol. Gen. Genet. 225:459-467.
Baumlein et al. (1992) Plant J. 2:233-239.
Belide et al. (2013) Plant Cell Tiss. Org. Cult. DOI 10.1007/s11240-013-0295-1.
Ben Saad et al. (2011) Transgenic Res 20: 1003-1018.
Benning et al (2008) Prog. Lipid Res. 47:381-389.
Benning et al (2009) J. Biol. Chem 284:17420-17427.
Bibikova et al. (2002) Genetics 161:1169-1175.
Bihmidine et al. (2015) BMC Plant Biology 15:186.
Bihmidine et al. (2016) Plant Signaling & Behaviour 11: e1117721.
Bligh and Dyer (1959) Canadian Journal of Biochemistry and Physiology 37:911-917.
Bourque (1995) Plant Sci. 105:125-149.
Boutilier et al. (2002) Plant Cell 14:1737-1749.
Bouvier-Nave et al. (2000) European Journal of Biochemistry/FEBS 267:85-96.
Bradford (1976) Anal. Biochem. 72:248-254.
Braun & Slewinski (2010), Plant Physiol 153: 1940.
Broothaerts et al. (2005) Nature 433:629-633.
Broun et al. (1998) Plant J. 13:201-210.
Browse et al. (1986) Biochem J 235: 25-31.
Buchanan-Wollaston (1994) Plant Physiol. 105:839-846.
Busk et al. (1997) Plant J. 11:1285-1295.
Cao et al. (2007) J. Lipid Res. 48:583-591.
Capuano et al. (2007) Biotechnol. Adv. 25:203-206.
Chen et al (2011) Plant Physiol. 155:851-865.
Chikwamba et al. (2003) Proc. Natl. Acad. Sci. U.S.A. 100:11127-11132.
Christie (1993) Advances in Lipid Methodology-Two, Oily Press, Dundee, pp195-213.
Chung et al. (2006) BMC Genomics 7:120.
Comai et al. (2004) Plant J 37: 778-786.
Cong et al. (2013) Science 339:819-823.
Corrado and Karali (2009) Biotechnol. Adv. 27:733-743.
Coutu et al. (2007) Transgenic Res. 16:771-781.
Dahlqvist et al. (2000), Proc. Natl. Acad. Sci. U.S.A. 97: 6487-6492.
Damaj et al., (2010) Planta 231:1439-1458.
Dandik and Aksoy (1998) Fuel Process Technol. 57: 81-92.
Dauk et al (2007) Plant Sci. 173:43-49.
Dulermo and Nicaud (2011) Metab. Eng. 13:482-491.
Durrett et al. (2008) Plant J. 54:593-607.
Dyer et al. (2002) Plant Physiol. 130:2027-2038.
Eastmond et al. (2006) Plant Cell 18: 665-675.
Ellerstrom et al. (1996) Plant Mol. Biol. 32:1019-1027.
Endalew et al. (2011) Biomass and Bioenergy 35:3787-3809.
Fan et al. (2013) Plant Cell 25: 3506-3518.
Fan et al. (2013) Plant Journal 76: 930-942.
Fan et al. (2014) Plant Cell 26: 4119-4134.
Fan et al. (2015) Plant Cell 27: 2941-2955.
FAO Animal Production and Health Proceedings (2002) Protein sources for the animal feed industry, Expert Consultation and Workshop, Bangkok.
Feeney et al. (2012) Plant Physiol 162: 1881-1896.
Finkelstein et al. (1998) Plant Cell 10:1043-1054.
Froissard et al. (2009) FEMS Yeast Res 9:428-438.
Gan (1995) Molecular characterization and genetic manipulation of plant senescence. PhD thesis. University of Wisconsin, Madison.
Gan and Amasino (1995) Science 270:1986-1988.
Gazzarrini et al. (2004) Dev. Cell 7:373-385.
Ghosal et al. (2007) Biochimica et Biophysica Acta 1771:1457-1463.
Ghosh et al. (2009) Plant Physiol. 151:869-881.
Gidda et al (2013) Plant Signaling Behav. 8:e27141.
Girijashankar and Swathisree, (2009) Physiol. Mol. Biol. Plants 15: 287-302.
Gong and Jiang (2011) Biotechnol. Lett. 33:1269-1284.
Gould et al. (1991) Plant Physiol. 95:426-434.
Greenwell et al. (2010) J. R. Soc. Interface 7:703-726.
Guan et al. (2015) Lipids 50:407-416.
Gurel et al. (2009) Plant Cell Rep. 28:429-444.
Gutierrez et al. (2013) BMC Biotechnol. 13: 40.
Hedrich et al. (2015) Curr Opin Plant Biol 25: 63-70.
Hershey and Stoner (1991) Plant Mol. Biol. 17:679-690.
Hinchee et al. (1988) Biotechnology 6:915-922.
Hom et al. (2007) Euphytica 153:27-34.
Hong et al. (2016). Progr Lipid Res 62:55-74.
Horn et al. (2013). Plant Physiol 162:1926-1936.
Horvath et al. (2000) Proc. Natl. Acad. Sci. U.S.A. 97:1914-1919.
Hsiao and Tzen (2011) Plant Physiol. Biochem. 49: 77-81.
Hu et al. (2012) Plant Physiol. 158:1944-1954.
Huang (1996) Plant Physiol. 110:1055-1061.
Huang et al. (2010). In Vitro Biology Meeting and IAPB 12th World Congress 2010, S93-S211
Huang and Huang (2016) Plant Physiol. 171: 1867-1878.
Ichihara et al (1988) Biochim. Biophys. Acta 958:125-129.
Ikeda et al. (2006) Pl Biotech J. 23: 153-161.
Iwabuchi et al. (2003) J. Biol. Chem. 278:4603-4610.
James et al. (2010) Proc. Natl. Acad. Sci. USA 107:17833-17838.
Jepson et al. (1994) Plant Mol. Biol. 26:1855-1866.
Jiang, et al. (2013) Nucleic Acids Research 41(20) e188.
Jolivet et al. (2014) Plant Physiol. Biochem. 42:501-509.
Jones et al. (1995) Plant Cell 7: 359-371.
Karmakar et al. (2010) Bioresource Technology 101:7201-7210.
Kelly et al. (2011) Plant Physiol. 157: 866-875.
Kelly et al (2013a) Plant Biotech. J. 11:355-361.
Kelly et al. (2013b) Plant Physiol. 162:1282-1289.
Kereszt et al. (2007) Nature Protocols 2:948-952.
Kim et al. (1996) Proc. Natl. Acad. Sci. USA 93:1156-1160.
Kim et al. (2016), Plant Physiol 171: 1951-1964.
Klemens et al. (2013) Plant Physiol 163: 1338-1352.
Koziel et al. (1996) Plant Mol. Biol. 32:393-405.
Kuhn et al. (2009) J. Biol. Chem. 284:34092-102.
Kunst et al. (1988) Proc. Natl. Acad. Sci. U.S.A. 85:4143-4147.
Kwong et al. (2003) Plant Cell 15:5-18.
Lacroix et al. (2008) Proc. Natl. Acad. Sci.U.S.A. 105: 15429-15434.
Laemmli (1970) Nature 227: 680-685.
Laibach et al. (2015). J. Biotechnol. 201: 15-27.
Lardizabal et al. (2008) Plant Physiol. 148: 89-96.
Larkin et al. (1996) Transgenic Res. 5:325-335.
Lebrun et al. (1987) Nucl. Acids Res. 15:4360.
Laux et al. (1996) Development 122: 87-96.
Lazo et al. (1991) Bio/Technology 9:963-967.
Lee et al. (1998) Science 280:915-918.
Lee et al., (2003) Proc. Natl. Acad. Sci. U.S.A. 100:2152-2158.
Li-Beisson et al (2013) The Arabidopsis Book, 2013.
Li et al. (1996) FEBS Lett. 379:117-121.
Li et al. (2006) Phytochemistry 67: 904-915.
Li et al. (2016). Inter. J. Agric. Biol. doi: 10.17957/IJAB/15.0075.
Lin et al. (2005) Plant Physiol. Biochem. 43:770-776.
Linder et al. (2005). FEMS Microbiol. Rev. 29: 877-896.
Liu and Godwin (2012). Plant Cell Reports 31, 999-1007.
Liu et al. (2010) Plant Physiol. Biochem. 48: 9-15.
Liu et al. (2012) J Exp Bot 63: 3727-3740.
Liu et al. (2014) BMC Plant Biol. 14: 73.
Liu et al. (2015). South African Journal of Botany 98, 157-160.
Liu et al. (2015b). Plant Cell 27:1512-1528.
Lotan et al. (1998) Cell 93: 1195-1205.
Lu et al (2007) J. Biol. Chem. 282: 35945-35953.
Lu et al. (2009) Proc Natl Acad of Sci USA 106:18837-18842.
Luerssen et al. (1998) Plant J. 15: 755-764.
Lui et al. (2009) J. Agric. Food Chem. 57: 2308-2313.
Ma et al. (2016) Plant Journal doi: 10.1111/tpj.13244.
MacEachran et al. (2010). Appl. Environ. Microbiol. 76: 7217-7225.
Maher and Bressler (2007) Bioresource Technology 98:2351-2368.
Matsuoka et al. (1994) Plant J. 6:311-319.
Matsuoka and Minami (1989) Eur. J. Biochem. 181: 593-598.
McCleary et al. (2013) J AOAC Int 93:221-233.
McCleary et al. (2015) Starch 67:860-883.
McElroy et al. (1990) Plant Cell 2: 163-171.
McKinley et al. (2016) Plant Journal: doi:10.1111/tpj.13269.
Meier et al. (1997) FEBS Lett. 415:91-95.
Millar and Waterhouse (2005). Funct Integr Genomics 5:129-135.
Miller (1984). Crop Sci 24:1224-1224.
Mizuno et al., (2016) Biotechnol Biofuels 9: 127.
Mojica et al. (2000) Mol Microbiol 36:244-246.
Mongrand et al. (1998) Phytochemistry 49:1049-1064.
Morelle et al., (2005). Eukaryot Cell 4:1308-1316.
Moreno-Perez (2012) PNAS 109:10107-10112.
Moyle and Birch (2013) Theor. Appl. Genet. 126:1775-1782.
Mu et al. (2008) Plant Physiol. 148:1042-1054.
Mudge et al., (2013) Plant Biotechnol. J. 11:502-509.
Murashige and Skoog (1962). Physiol Plant 15:473-497.
Murphy et al. (2012). Protoplasma 249:541-585.
Naim et al. (2012) PLoS One 7: e52717.
Nakamura et al., (2005). J Biol Chem 280:7469-7476.
Needleman and Wunsch (1970) J. Mol Biol. 45: 443-453.
Nilsson et al. (2012) Physiol. Plantarum 144: 35-47.
Nishida et al (1993) Plant Mol. Biol. 21:267-277.
Nomura et al. (2000) Plant Mol. Biol. 44: 99-106.
Ohlrogge and Browse (1995) Plant Cell 7: 957-970.
Padidam (2003) Curr. Opin. Plant Biol. 6:169-77.
Padidam et al. (2003) Transgenic Res. 12:101-9.
Parthibane et al. (2012a) J. Biol. Chem. 287:1946-1965.
Parthibane et al. (2012b) Plant Physiol. 159:95-104.
Pasquinelli et al. (2005). Curr. Opin. Genet. Develop. 15:200-205.
Pearson and Lipman (1988) Proc. Natl. Acad. Sci. USA 85:2444-2448.
Perez-Vich et al. (1998) J.A.O.C.S. 75:547-555.
Perrin et al. (2000) Mol. Breed. 6:345-352.
Petrie et al. (2012) PLOS One 7: e35214.
Phillips et al. (2002) Journal of Food Composition and Analysis 12:123-142.
Pokotylo et al., 2013. Progr Lipid Res. 52:62-79.
Potenza et al. (2004) In Vitro Cell Dev. Biol. Plant 40:1-22.
Prosky et al. (1985) J AOAC Chem 68:677-679.
Qazi et al. (2012) Journal of Plant Physiology 169: 605-613.
Qiu et al. (2001) J. Biol. Chem. 276:31561-3156.
Robson et al. (2004) Plant Biotechnol J 2:101-112.
Rossell and Pritchard (1991) Analysis of Oilseeds, Fats and Fatty Foods. Elsevier Roston et al (2012) J. Biol. Chem. 287:21406-21415.
Ruuska et al. (2002) Plant Cell 14:1191-1206.
Saha et al. (2006) Plant Physiol. 141:1533-1543.
Sanjaya et al. (2011) Plant Biotechnol J 9:874-883.
Santos-Mendoza et al. (2005) FEBS Lett. 579:4666-4670.
Santos-Mendoza et al. (2008) Plant J. 54:608-620.
Schneider et al. (2012) Plant Biol 14: 325-336.
Schnurr et al. (2002) Plant Physiol 129:1700-1709.
Scott et al. (2010) Plant Biotechnol. J. 8:912-27.
Shaw et al. (1959) J Soil Sci 10:316-326.
Shen et al. (2010) Plant Phys. 153: 980-987.
Shen et al. (2014). Biochem. Biophys. Res. Comm. 448: 365-371.
Semwal et al. (2011) Bioresource Technology 102:2151-2161.
Senior (1998) Biotech. Genet. Engin. Revs. 15:79-119.
Shen et al. (2010) Plant Physiol. 153:980-987.
Shiina et al. (1997) Plant Physiol. 115:477-483.
Shimada and Hara-Nishimura (2010) Biol. Pharm. Bull. 33:360-363.
Shimada et al. (2014) Plant Physiol. 164:105-118.
Shockey et al. (2002) Plant Physiol 129:1710-1722.
Singh et al., (2013). PLoS One 8, e62494.
Slade and Knauf (2005) Transgenic Res. 14: 109-115.
Slocombe et al. (2009) Plant Biotechnol. J. 7: 694-703.
Smith et al. (2000) Nature 407:319-320.
Somerville et al. (2000) Lipids. In BB Buchanan, W Gruissem, RL Jones, eds, Biochemisty and Molecular Biology of Plants. American Society of Plant Physiologists, Rockville, MD, pp 456-527.
Srinivasan et al. (2007) Planta 225:341-51.
Stalker et al. 1988 Science 242: 419-423.
Stone et al. (2001) Proc. Natl. Acad. Sci. U.S.A.98: 11806-11811.
Stone et al. (2008) Proc. Natl. Acad. Sci. U.S.A.105: 3151-3156.
Tai et al. (2002). Biosci. Biotechnol. Biochem. 66: 2146-2153.
Tan et al. (2011) Plant Physiol. 156:1577-1588.
Taylor (1997) The Plant Cell 9:1245-1249.
Thillet et al. (1988) J. Biol. Chem 263:12500-12508.
Tingay et al. (1997) Plant J. 11:1369-1376.
Titball 1993. Microbiol Rev 57:347-366.
To et al. (2012) Plant Cell 24:5007-5023.
Ulmasov et al. (1995) Plant Physiol. 108:919-927.
van de Loo et al. (1995) Proc Natl Acad Sci U S A. 92:6743-6747.
Vanhercke et al. (2013) FEBS Letters 587:364-369.
Vanhercke et al. (2014). Plant Biotech. J. 12:231-239.
Vieler et al. (2012) Plant Physiol. 158:1562-1569.
Voinnet et al. (2003) Plant J. 33:949-956.
Wang and Benning (2012) Plant J 70:614-623.
Wang et al., (2001). Annu Rev Plant Physiol Plant Mol Biol 52:211-231.
Wang et al. (2002) Plant J 32:831-843.
Wang (2005). Plant Physiol 139:566-573.
Waterhouse et al. (1998). Proc. Natl. Acad. Sci. U.S.A. 95:13959-13964.
Weissbach and Weissbach, (1989) Methods for Plant Mol Biol, Academic Press.
Weissbach et al., In: Methods for Plant Molecular Biology, Academic Press, San Diego, Calif., (1988).
Winichayakul et al. (2013) Plant Physiol. 162:626-639.
Wood et al. (2009) Plant Biotech. J. 7: 914-924.
Wormit et al. (2006) Plant Cell 18: 3476-3490.
Wright et al. (2006) Methods Mol Biol. 343:120-135.
Wu et al. (2014) In Vitro Cellular and Dev. Biol.-Plant 50:9-18.
Xie et al. (2014) Mol. Plant 7:923-926.
Xu et al (2010) Plant and Cell Physiol. 51:1019-1028.
Xu et al (2005) Plant Cell 17:3094-3110.
Xu et al (2008) Plant Cell 20:2190-2204.
Yamagishi et al. (2005) Pl Physiol 139: 163-173.
Yamasaki et al. (2004) Plant Cell 16:3448-3459.
Yang et al. (2003) Planta 216:597-603.
Yang et al. (2010) Proc. Natl. Acad. Sci. U.S.A.107:12040-12045.
Yen et al. (2002) Proc. Natl. Acad. Sci. U.S.A. 99:8512-8517.
Yen et al. (2005) J. Lipid Res. 46: 1502-1511.
Yokoyama et al. (1994) Mol Gen Genet 244: 15-22.
Zale et al. (2016), Plant Biotech J. 14: 661-669.
Zheng et al. (2009) Pl Physiol 21: 2563-2577.
Zienkiewicz et al. (2017) Biotechnology for Biofuels 10 doi:http://dx.doi.org/10.1186/ s13068-016-0686-8
Zolman et al (2001) Plant Physiol. 127:1266-1274.
Zulu et al. (2017) Biotechnology for Biofuels, 10 doi:https://doi.org/10.1186/s13068-017-0874-1.
--->
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Commonwealth Scientific and Industrial Research
Organisation
<120> Растения с модифицированными признаками
<130> 526280
<150> AU 2017902756
<151> 2017-07-13
<150> PCT/AU2017/050012
<151> 2017-01-06
<150> AU 2016903541
<151> 2016-09-02
<150> AU 2016903577
<151> 2016-09-06
<150> AU 2016904611
<151> 2016-11-11
<160> 323
<170> PatentIn версии 3.5
<210> 1
<211> 520
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 1
Met Ala Ile Leu Asp Ser Ala Gly Val Thr Thr Val Thr Glu Asn Gly
1 5 10 15
Gly Gly Glu Phe Val Asp Leu Asp Arg Leu Arg Arg Arg Lys Ser Arg
20 25 30
Ser Asp Ser Ser Asn Gly Leu Leu Leu Ser Gly Ser Asp Asn Asn Ser
35 40 45
Pro Ser Asp Asp Val Gly Ala Pro Ala Asp Val Arg Asp Arg Ile Asp
50 55 60
Ser Val Val Asn Asp Asp Ala Gln Gly Thr Ala Asn Leu Ala Gly Asp
65 70 75 80
Asn Asn Gly Gly Gly Asp Asn Asn Gly Gly Gly Arg Gly Gly Gly Glu
85 90 95
Gly Arg Gly Asn Ala Asp Ala Thr Phe Thr Tyr Arg Pro Ser Val Pro
100 105 110
Ala His Arg Arg Ala Arg Glu Ser Pro Leu Ser Ser Asp Ala Ile Phe
115 120 125
Lys Gln Ser His Ala Gly Leu Phe Asn Leu Cys Val Val Val Leu Ile
130 135 140
Ala Val Asn Ser Arg Leu Ile Ile Glu Asn Leu Met Lys Tyr Gly Trp
145 150 155 160
Leu Ile Arg Thr Asp Phe Trp Phe Ser Ser Arg Ser Leu Arg Asp Trp
165 170 175
Pro Leu Phe Met Cys Cys Ile Ser Leu Ser Ile Phe Pro Leu Ala Ala
180 185 190
Phe Thr Val Glu Lys Leu Val Leu Gln Lys Tyr Ile Ser Glu Pro Val
195 200 205
Val Ile Phe Leu His Ile Ile Ile Thr Met Thr Glu Val Leu Tyr Pro
210 215 220
Val Tyr Val Thr Leu Arg Cys Asp Ser Ala Phe Leu Ser Gly Val Thr
225 230 235 240
Leu Met Leu Leu Thr Cys Ile Val Trp Leu Lys Leu Val Ser Tyr Ala
245 250 255
His Thr Ser Tyr Asp Ile Arg Ser Leu Ala Asn Ala Ala Asp Lys Ala
260 265 270
Asn Pro Glu Val Ser Tyr Tyr Val Ser Leu Lys Ser Leu Ala Tyr Phe
275 280 285
Met Val Ala Pro Thr Leu Cys Tyr Gln Pro Ser Tyr Pro Arg Ser Ala
290 295 300
Cys Ile Arg Lys Gly Trp Val Ala Arg Gln Phe Ala Lys Leu Val Ile
305 310 315 320
Phe Thr Gly Phe Met Gly Phe Ile Ile Glu Gln Tyr Ile Asn Pro Ile
325 330 335
Val Arg Asn Ser Lys His Pro Leu Lys Gly Asp Leu Leu Tyr Ala Ile
340 345 350
Glu Arg Val Leu Lys Leu Ser Val Pro Asn Leu Tyr Val Trp Leu Cys
355 360 365
Met Phe Tyr Cys Phe Phe His Leu Trp Leu Asn Ile Leu Ala Glu Leu
370 375 380
Leu Cys Phe Gly Asp Arg Glu Phe Tyr Lys Asp Trp Trp Asn Ala Lys
385 390 395 400
Ser Val Gly Asp Tyr Trp Arg Met Trp Asn Met Pro Val His Lys Trp
405 410 415
Met Val Arg His Ile Tyr Phe Pro Cys Leu Arg Ser Lys Ile Pro Lys
420 425 430
Thr Leu Ala Ile Ile Ile Ala Phe Leu Val Ser Ala Val Phe His Glu
435 440 445
Leu Cys Ile Ala Val Pro Cys Arg Leu Phe Lys Leu Trp Ala Phe Leu
450 455 460
Gly Ile Met Phe Gln Val Pro Leu Val Phe Ile Thr Asn Tyr Leu Gln
465 470 475 480
Glu Arg Phe Gly Ser Thr Val Gly Asn Met Ile Phe Trp Phe Ile Phe
485 490 495
Cys Ile Phe Gly Gln Pro Met Cys Val Leu Leu Tyr Tyr His Asp Leu
500 505 510
Met Asn Arg Lys Gly Ser Met Ser
515 520
<210> 2
<211> 314
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 2
Met Gly Gly Ser Arg Glu Phe Arg Ala Glu Glu His Ser Asn Gln Phe
1 5 10 15
His Ser Ile Ile Ala Met Ala Ile Trp Leu Gly Ala Ile His Phe Asn
20 25 30
Val Ala Leu Val Leu Cys Ser Leu Ile Phe Leu Pro Pro Ser Leu Ser
35 40 45
Leu Met Val Leu Gly Leu Leu Ser Leu Phe Ile Phe Ile Pro Ile Asp
50 55 60
His Arg Ser Lys Tyr Gly Arg Lys Leu Ala Arg Tyr Ile Cys Lys His
65 70 75 80
Ala Cys Asn Tyr Phe Pro Val Ser Leu Tyr Val Glu Asp Tyr Glu Ala
85 90 95
Phe Gln Pro Asn Arg Ala Tyr Val Phe Gly Tyr Glu Pro His Ser Val
100 105 110
Leu Pro Ile Gly Val Val Ala Leu Cys Asp Leu Thr Gly Phe Met Pro
115 120 125
Ile Pro Asn Ile Lys Val Leu Ala Ser Ser Ala Ile Phe Tyr Thr Pro
130 135 140
Phe Leu Arg His Ile Trp Thr Trp Leu Gly Leu Thr Ala Ala Ser Arg
145 150 155 160
Lys Asn Phe Thr Ser Leu Leu Asp Ser Gly Tyr Ser Cys Val Leu Val
165 170 175
Pro Gly Gly Val Gln Glu Thr Phe His Met Gln His Asp Ala Glu Asn
180 185 190
Val Phe Leu Ser Arg Arg Arg Gly Phe Val Arg Ile Ala Met Glu Gln
195 200 205
Gly Ser Pro Leu Val Pro Val Phe Cys Phe Gly Gln Ala Arg Val Tyr
210 215 220
Lys Trp Trp Lys Pro Asp Cys Asp Leu Tyr Leu Lys Leu Ser Arg Ala
225 230 235 240
Ile Arg Phe Thr Pro Ile Cys Phe Trp Gly Val Phe Gly Ser Pro Leu
245 250 255
Pro Cys Arg Gln Pro Met His Val Val Val Gly Lys Pro Ile Glu Val
260 265 270
Thr Lys Thr Leu Lys Pro Thr Asp Glu Glu Ile Ala Lys Phe His Gly
275 280 285
Gln Tyr Val Glu Ala Leu Arg Asp Leu Phe Glu Arg His Lys Ser Arg
290 295 300
Val Gly Tyr Asp Leu Glu Leu Lys Ile Leu
305 310
<210> 3
<211> 340
<212> БЕЛОК
<213> Ricinus communis
<400> 3
Met Gly Glu Glu Ala Asn His Asn Asn Asn Asn Asn Asn Ile Asn Ser
1 5 10 15
Asn Asp Glu Lys Asn Glu Glu Lys Ser Asn Tyr Thr Val Val Asn Ser
20 25 30
Arg Glu Leu Tyr Pro Thr Asn Ile Phe His Ala Leu Leu Ala Leu Ser
35 40 45
Ile Trp Ile Gly Ser Ile His Phe Asn Leu Phe Leu Leu Phe Ile Ser
50 55 60
Tyr Leu Phe Leu Ser Phe Pro Thr Phe Leu Leu Ile Val Gly Phe Phe
65 70 75 80
Val Val Leu Met Phe Ile Pro Ile Asp Glu His Ser Lys Leu Gly Arg
85 90 95
Arg Leu Cys Arg Tyr Val Cys Arg His Ala Cys Ser His Phe Pro Val
100 105 110
Thr Leu His Val Glu Asp Met Asn Ala Phe His Ser Asp Arg Ala Tyr
115 120 125
Val Phe Gly Tyr Glu Pro His Ser Val Phe Pro Leu Gly Val Ser Val
130 135 140
Leu Ser Asp His Phe Ala Val Leu Pro Leu Pro Lys Met Lys Val Leu
145 150 155 160
Ala Ser Asn Ala Val Phe Arg Thr Pro Val Leu Arg His Ile Trp Thr
165 170 175
Trp Cys Gly Leu Thr Ser Ala Thr Lys Lys Asn Phe Thr Ala Leu Leu
180 185 190
Ala Ser Gly Tyr Ser Cys Ile Val Ile Pro Gly Gly Val Gln Glu Thr
195 200 205
Phe Tyr Met Lys His Gly Ser Glu Ile Ala Phe Leu Lys Ala Arg Arg
210 215 220
Gly Phe Val Arg Val Ala Met Glu Met Gly Lys Pro Leu Val Pro Val
225 230 235 240
Phe Cys Phe Gly Gln Ser Asn Val Tyr Lys Trp Trp Lys Pro Asp Gly
245 250 255
Glu Leu Phe Met Lys Ile Ala Arg Ala Ile Lys Phe Ser Pro Ile Val
260 265 270
Phe Trp Gly Val Leu Gly Ser His Leu Pro Leu Gln Arg Pro Met His
275 280 285
Val Val Val Gly Lys Pro Ile Glu Val Lys Gln Asn Pro Gln Pro Thr
290 295 300
Val Glu Glu Val Ser Glu Val Gln Gly Gln Phe Val Ala Ala Leu Lys
305 310 315 320
Asp Leu Phe Glu Arg His Lys Ala Arg Val Gly Tyr Ala Asp Leu Thr
325 330 335
Leu Glu Ile Leu
340
<210> 4
<211> 322
<212> БЕЛОК
<213> Vernicia fordii
<400> 4
Met Gly Met Val Glu Val Lys Asn Glu Glu Glu Val Thr Ile Phe Lys
1 5 10 15
Ser Gly Glu Ile Tyr Pro Thr Asn Ile Phe Gln Ser Val Leu Ala Leu
20 25 30
Ala Ile Trp Leu Gly Ser Phe His Phe Ile Leu Phe Leu Val Ser Ser
35 40 45
Ser Ile Phe Leu Pro Phe Ser Lys Phe Leu Leu Val Ile Gly Leu Leu
50 55 60
Leu Phe Phe Met Val Ile Pro Ile Asn Asp Arg Ser Lys Leu Gly Gln
65 70 75 80
Cys Leu Phe Ser Tyr Ile Ser Arg His Val Cys Ser Tyr Phe Pro Ile
85 90 95
Thr Leu His Val Glu Asp Ile Asn Ala Phe Arg Ser Asp Arg Ala Tyr
100 105 110
Val Phe Gly Tyr Glu Pro His Ser Val Phe Pro Ile Gly Val Met Ile
115 120 125
Leu Ser Leu Gly Leu Ile Pro Leu Pro Asn Ile Lys Phe Leu Ala Ser
130 135 140
Ser Ala Val Phe Tyr Thr Pro Phe Leu Arg His Ile Trp Ser Trp Cys
145 150 155 160
Gly Leu Thr Pro Ala Thr Arg Lys Asn Phe Val Ser Leu Leu Ser Ser
165 170 175
Gly Tyr Ser Cys Ile Leu Val Pro Gly Gly Val Gln Glu Thr Phe Tyr
180 185 190
Met Lys Gln Asp Ser Glu Ile Ala Phe Leu Lys Ala Arg Arg Gly Phe
195 200 205
Ile Arg Ile Ala Met Gln Thr Gly Thr Pro Leu Val Pro Val Phe Cys
210 215 220
Phe Gly Gln Met His Thr Phe Lys Trp Trp Lys Pro Asp Gly Glu Leu
225 230 235 240
Phe Met Lys Ile Ala Arg Ala Ile Lys Phe Thr Pro Thr Ile Phe Trp
245 250 255
Gly Val Leu Gly Thr Pro Leu Pro Phe Lys Asn Pro Met His Val Val
260 265 270
Val Gly Arg Pro Ile Glu Val Lys Gln Asn Pro Gln Pro Thr Ala Glu
275 280 285
Glu Val Ala Glu Val Gln Arg Glu Phe Ile Ala Ser Leu Lys Asn Leu
290 295 300
Phe Glu Arg His Lys Ala Arg Val Gly Tyr Ser Asp Leu Lys Leu Glu
305 310 315 320
Ile Phe
<210> 5
<211> 355
<212> БЕЛОК
<213> Mortierella ramanniana
<400> 5
Met Ala Ser Lys Asp Gln His Leu Gln Gln Lys Val Lys His Thr Leu
1 5 10 15
Glu Ala Ile Pro Ser Pro Arg Tyr Ala Pro Leu Arg Val Pro Leu Arg
20 25 30
Arg Arg Leu Gln Thr Leu Ala Val Leu Leu Trp Cys Ser Met Met Ser
35 40 45
Ile Cys Met Phe Ile Phe Phe Phe Leu Cys Ser Ile Pro Val Leu Leu
50 55 60
Trp Phe Pro Ile Ile Leu Tyr Leu Thr Trp Ile Leu Val Trp Asp Lys
65 70 75 80
Ala Pro Glu Asn Gly Gly Arg Pro Ile Arg Trp Leu Arg Asn Ala Ala
85 90 95
Trp Trp Lys Leu Phe Ala Gly Tyr Phe Pro Ala His Val Ile Lys Glu
100 105 110
Ala Asp Leu Asp Pro Ser Lys Asn Tyr Ile Phe Gly Tyr His Pro His
115 120 125
Gly Ile Ile Ser Met Gly Ser Phe Cys Thr Phe Ser Thr Asn Ala Thr
130 135 140
Gly Phe Asp Asp Leu Phe Pro Gly Ile Arg Pro Ser Leu Leu Thr Leu
145 150 155 160
Thr Ser Asn Phe Asn Ile Pro Leu Tyr Arg Asp Tyr Leu Met Ala Cys
165 170 175
Gly Leu Cys Ser Val Ser Lys Thr Ser Cys Gln Asn Ile Leu Thr Lys
180 185 190
Gly Gly Pro Gly Arg Ser Ile Ala Ile Val Val Gly Gly Ala Ser Glu
195 200 205
Ser Leu Asn Ala Arg Pro Gly Val Met Asp Leu Val Leu Lys Arg Arg
210 215 220
Phe Gly Phe Ile Lys Ile Ala Val Gln Thr Gly Ala Ser Leu Val Pro
225 230 235 240
Thr Ile Ser Phe Gly Glu Asn Glu Leu Tyr Glu Gln Ile Glu Ser Asn
245 250 255
Glu Asn Ser Lys Leu His Arg Trp Gln Lys Lys Ile Gln His Ala Leu
260 265 270
Gly Phe Thr Met Pro Leu Phe His Gly Arg Gly Val Phe Asn Tyr Asp
275 280 285
Phe Gly Leu Leu Pro His Arg His Pro Ile Tyr Thr Ile Val Gly Lys
290 295 300
Pro Ile Pro Val Pro Ser Ile Lys Tyr Gly Gln Thr Lys Asp Glu Ile
305 310 315 320
Ile Arg Glu Leu His Asp Ser Tyr Met His Ala Val Gln Asp Leu Tyr
325 330 335
Asp Arg Tyr Lys Asp Ile Tyr Ala Lys Asp Arg Val Lys Glu Leu Glu
340 345 350
Phe Val Glu
355
<210> 6
<211> 388
<212> БЕЛОК
<213> Homo sapiens
<400> 6
Met Lys Thr Leu Ile Ala Ala Tyr Ser Gly Val Leu Arg Gly Glu Arg
1 5 10 15
Gln Ala Glu Ala Asp Arg Ser Gln Arg Ser His Gly Gly Pro Ala Leu
20 25 30
Ser Arg Glu Gly Ser Gly Arg Trp Gly Thr Gly Ser Ser Ile Leu Ser
35 40 45
Ala Leu Gln Asp Leu Phe Ser Val Thr Trp Leu Asn Arg Ser Lys Val
50 55 60
Glu Lys Gln Leu Gln Val Ile Ser Val Leu Gln Trp Val Leu Ser Phe
65 70 75 80
Leu Val Leu Gly Val Ala Cys Ser Ala Ile Leu Met Tyr Ile Phe Cys
85 90 95
Thr Asp Cys Trp Leu Ile Ala Val Leu Tyr Phe Thr Trp Leu Val Phe
100 105 110
Asp Trp Asn Thr Pro Lys Lys Gly Gly Arg Arg Ser Gln Trp Val Arg
115 120 125
Asn Trp Ala Val Trp Arg Tyr Phe Arg Asp Tyr Phe Pro Ile Gln Leu
130 135 140
Val Lys Thr His Asn Leu Leu Thr Thr Arg Asn Tyr Ile Phe Gly Tyr
145 150 155 160
His Pro His Gly Ile Met Gly Leu Gly Ala Phe Cys Asn Phe Ser Thr
165 170 175
Glu Ala Thr Glu Val Ser Lys Lys Phe Pro Gly Ile Arg Pro Tyr Leu
180 185 190
Ala Thr Leu Ala Gly Asn Phe Arg Met Pro Val Leu Arg Glu Tyr Leu
195 200 205
Met Ser Gly Gly Ile Cys Pro Val Ser Arg Asp Thr Ile Asp Tyr Leu
210 215 220
Leu Ser Lys Asn Gly Ser Gly Asn Ala Ile Ile Ile Val Val Gly Gly
225 230 235 240
Ala Ala Glu Ser Leu Ser Ser Met Pro Gly Lys Asn Ala Val Thr Leu
245 250 255
Arg Asn Arg Lys Gly Phe Val Lys Leu Ala Leu Arg His Gly Ala Asp
260 265 270
Leu Val Pro Ile Tyr Ser Phe Gly Glu Asn Glu Val Tyr Lys Gln Val
275 280 285
Ile Phe Glu Glu Gly Ser Trp Gly Arg Trp Val Gln Lys Lys Phe Gln
290 295 300
Lys Tyr Ile Gly Phe Ala Pro Cys Ile Phe His Gly Arg Gly Leu Phe
305 310 315 320
Ser Ser Asp Thr Trp Gly Leu Val Pro Tyr Ser Lys Pro Ile Thr Thr
325 330 335
Val Val Gly Glu Pro Ile Thr Ile Pro Lys Leu Glu His Pro Thr Gln
340 345 350
Gln Asp Ile Asp Leu Tyr His Thr Met Tyr Met Glu Ala Leu Val Lys
355 360 365
Leu Phe Asp Lys His Lys Thr Lys Phe Gly Leu Pro Glu Thr Glu Val
370 375 380
Leu Glu Val Asn
385
<210> 7
<211> 328
<212> БЕЛОК
<213> Homo sapiens
<400> 7
Met Ala His Ser Lys Gln Pro Ser His Phe Gln Ser Leu Met Leu Leu
1 5 10 15
Gln Trp Pro Leu Ser Tyr Leu Ala Ile Phe Trp Ile Leu Gln Pro Leu
20 25 30
Phe Val Tyr Leu Leu Phe Thr Ser Leu Trp Pro Leu Pro Val Leu Tyr
35 40 45
Phe Ala Trp Leu Phe Leu Asp Trp Lys Thr Pro Glu Arg Gly Gly Arg
50 55 60
Arg Ser Ala Trp Val Arg Asn Trp Cys Val Trp Thr His Ile Arg Asp
65 70 75 80
Tyr Phe Pro Ile Thr Ile Leu Lys Thr Lys Asp Leu Ser Pro Glu His
85 90 95
Asn Tyr Leu Met Gly Val His Pro His Gly Leu Leu Thr Phe Gly Ala
100 105 110
Phe Cys Asn Phe Cys Thr Glu Ala Thr Gly Phe Ser Lys Thr Phe Pro
115 120 125
Gly Ile Thr Pro His Leu Ala Thr Leu Ser Trp Phe Phe Lys Ile Pro
130 135 140
Phe Val Arg Glu Tyr Leu Met Ala Lys Gly Val Cys Ser Val Ser Gln
145 150 155 160
Pro Ala Ile Asn Tyr Leu Leu Ser His Gly Thr Gly Asn Leu Val Gly
165 170 175
Ile Val Val Gly Gly Val Gly Glu Ala Leu Gln Ser Val Pro Asn Thr
180 185 190
Thr Thr Leu Ile Leu Gln Lys Arg Lys Gly Phe Val Arg Thr Ala Leu
195 200 205
Gln His Gly Ala His Leu Val Pro Thr Phe Thr Phe Gly Glu Thr Glu
210 215 220
Val Tyr Asp Gln Val Leu Phe His Lys Asp Ser Arg Met Tyr Lys Phe
225 230 235 240
Gln Ser Cys Phe Arg Arg Ile Phe Gly Phe Tyr Cys Cys Val Phe Tyr
245 250 255
Gly Gln Ser Phe Cys Gln Gly Ser Thr Gly Leu Leu Pro Tyr Ser Arg
260 265 270
Pro Ile Val Thr Val Val Gly Glu Pro Leu Pro Leu Pro Gln Ile Glu
275 280 285
Lys Pro Ser Gln Glu Met Val Asp Lys Tyr His Ala Leu Tyr Met Asp
290 295 300
Ala Leu His Lys Leu Phe Asp Gln His Lys Thr His Tyr Gly Cys Ser
305 310 315 320
Glu Thr Gln Lys Leu Phe Phe Leu
325
<210> 8
<211> 361
<212> БЕЛОК
<213> Bos taurus
<400> 8
Met Lys Thr Leu Ile Ala Ala Tyr Ser Gly Val Leu Arg Gly Thr Gly
1 5 10 15
Ser Ser Ile Leu Ser Ala Leu Gln Asp Leu Phe Ser Val Thr Trp Leu
20 25 30
Asn Arg Ala Lys Val Glu Lys Gln Leu Gln Val Ile Ser Val Leu Gln
35 40 45
Trp Val Leu Ser Phe Leu Val Leu Gly Val Ala Cys Ser Val Ile Leu
50 55 60
Met Tyr Thr Phe Cys Thr Asp Cys Trp Leu Ile Ala Val Leu Tyr Phe
65 70 75 80
Thr Trp Leu Val Phe Asp Trp Asn Thr Pro Lys Lys Gly Gly Arg Arg
85 90 95
Ser Gln Trp Val Arg Asn Trp Ala Val Trp Arg Tyr Phe Arg Asp Tyr
100 105 110
Phe Pro Ile Gln Leu Val Lys Thr His Asn Leu Leu Thr Ser Arg Asn
115 120 125
Tyr Ile Phe Gly Tyr His Pro His Gly Ile Met Gly Leu Gly Ala Phe
130 135 140
Cys Asn Phe Ser Thr Glu Ala Thr Glu Val Ser Lys Lys Phe Pro Gly
145 150 155 160
Ile Arg Pro Tyr Leu Ala Thr Leu Ala Gly Asn Phe Arg Met Pro Val
165 170 175
Leu Arg Glu Tyr Leu Met Ser Gly Gly Ile Cys Pro Val Asn Arg Asp
180 185 190
Thr Ile Asp Tyr Leu Leu Ser Lys Asn Gly Ser Gly Asn Ala Ile Ile
195 200 205
Ile Val Val Gly Gly Ala Ala Glu Ser Leu Ser Ser Met Pro Gly Lys
210 215 220
Asn Ala Val Thr Leu Arg Asn Arg Lys Gly Phe Val Lys Leu Ala Leu
225 230 235 240
Arg His Gly Ala Asp Leu Val Pro Thr Tyr Ser Phe Gly Glu Asn Glu
245 250 255
Val Tyr Lys Gln Val Ile Phe Glu Glu Gly Ser Trp Gly Arg Trp Val
260 265 270
Gln Lys Lys Phe Gln Lys Tyr Ile Gly Phe Ala Pro Cys Ile Phe His
275 280 285
Gly Arg Gly Leu Phe Ser Ser Asp Thr Trp Gly Leu Val Pro Tyr Ser
290 295 300
Lys Pro Ile Thr Thr Val Val Gly Glu Pro Ile Thr Ile Pro Arg Leu
305 310 315 320
Glu Arg Pro Thr Gln Gln Asp Ile Asp Leu Tyr His Ala Met Tyr Val
325 330 335
Gln Ala Leu Val Lys Leu Phe Asp Gln His Lys Thr Lys Phe Gly Leu
340 345 350
Pro Glu Thr Glu Val Leu Glu Val Asn
355 360
<210> 9
<211> 388
<212> БЕЛОК
<213> Mus musculus
<400> 9
Met Lys Thr Leu Ile Ala Ala Tyr Ser Gly Val Leu Arg Gly Glu Arg
1 5 10 15
Arg Ala Glu Ala Ala Arg Ser Glu Asn Lys Asn Lys Gly Ser Ala Leu
20 25 30
Ser Arg Glu Gly Ser Gly Arg Trp Gly Thr Gly Ser Ser Ile Leu Ser
35 40 45
Ala Leu Gln Asp Ile Phe Ser Val Thr Trp Leu Asn Arg Ser Lys Val
50 55 60
Glu Lys Gln Leu Gln Val Ile Ser Val Leu Gln Trp Val Leu Ser Phe
65 70 75 80
Leu Val Leu Gly Val Ala Cys Ser Val Ile Leu Met Tyr Thr Phe Cys
85 90 95
Thr Asp Cys Trp Leu Ile Ala Val Leu Tyr Phe Thr Trp Leu Ala Phe
100 105 110
Asp Trp Asn Thr Pro Lys Lys Gly Gly Arg Arg Ser Gln Trp Val Arg
115 120 125
Asn Trp Ala Val Trp Arg Tyr Phe Arg Asp Tyr Phe Pro Ile Gln Leu
130 135 140
Val Lys Thr His Asn Leu Leu Thr Thr Arg Asn Tyr Ile Phe Gly Tyr
145 150 155 160
His Pro His Gly Ile Met Gly Leu Gly Ala Phe Cys Asn Phe Ser Thr
165 170 175
Glu Ala Thr Glu Val Ser Lys Lys Phe Pro Gly Ile Arg Pro Tyr Leu
180 185 190
Ala Thr Leu Ala Gly Asn Phe Arg Met Pro Val Leu Arg Glu Tyr Leu
195 200 205
Met Ser Gly Gly Ile Cys Pro Val Asn Arg Asp Thr Ile Asp Tyr Leu
210 215 220
Leu Ser Lys Asn Gly Ser Gly Asn Ala Ile Ile Ile Val Val Gly Gly
225 230 235 240
Ala Ala Glu Ser Leu Ser Ser Met Pro Gly Lys Asn Ala Val Thr Leu
245 250 255
Lys Asn Arg Lys Gly Phe Val Lys Leu Ala Leu Arg His Gly Ala Asp
260 265 270
Leu Val Pro Thr Tyr Ser Phe Gly Glu Asn Glu Val Tyr Lys Gln Val
275 280 285
Ile Phe Glu Glu Gly Ser Trp Gly Arg Trp Val Gln Lys Lys Phe Gln
290 295 300
Lys Tyr Ile Gly Phe Ala Pro Cys Ile Phe His Gly Arg Gly Leu Phe
305 310 315 320
Ser Ser Asp Thr Trp Gly Leu Val Pro Tyr Ser Lys Pro Ile Thr Thr
325 330 335
Val Val Gly Glu Pro Ile Thr Val Pro Lys Leu Glu His Pro Thr Gln
340 345 350
Lys Asp Ile Asp Leu Tyr His Ala Met Tyr Met Glu Ala Leu Val Lys
355 360 365
Leu Phe Asp Asn His Lys Thr Lys Phe Gly Leu Pro Glu Thr Glu Val
370 375 380
Leu Glu Val Asn
385
<210> 10
<211> 3
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> консервативная последовательность
<400> 10
Tyr Phe Pro
1
<210> 11
<211> 4
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> консервативная последовательность
<400> 11
His Pro His Gly
1
<210> 12
<211> 4
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> консервативная последовательность
<400> 12
Glu Pro His Ser
1
<210> 13
<211> 24
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> консервативная последовательность
<220>
<221> X
<222> (2)..(2)
<223> любая аминокислота
<220>
<221> X
<222> (5)..(5)
<223> любая аминокислота
<220>
<221> X
<222> (6)..(6)
<223> лизин (K) или аргинин (R)
<220>
<221> X
<222> (7)..(7)
<223> любая аминокислота
<220>
<221> X
<222> (9)..(11)
<223> любая аминокислота
<220>
<221> X
<222> (13)..(15)
<223> любая аминокислота
<220>
<221> X
<222> (16)..(16)
<223> лейцин (L) или валин (V)
<220>
<221> X
<222> (19)..(21)
<223> любая аминокислота
<220>
<221> X
<222> (24)..(24)
<223> глутаминовая кислота (E) или глутамин (Q)
<400> 13
Arg Xaa Gly Phe Xaa Xaa Xaa Ala Xaa Xaa Xaa Gly Xaa Xaa Xaa Xaa
1 5 10 15
Val Pro Xaa Xaa Xaa Phe Gly Xaa
20
<210> 14
<211> 8
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> консервативная последовательность
<220>
<221> X
<222> (3)..(3)
<223> любая аминокислота
<220>
<221> X
<222> (5)..(7)
<223> любая аминокислота
<400> 14
Phe Leu Xaa Leu Xaa Xaa Xaa Asn
1 5
<210> 15
<211> 118
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> консервативная последовательность
<400> 15
Ala Leu Val Val Ala Asn His Gln Ser Phe Leu Asp Pro Leu Val Leu
1 5 10 15
Ser Ala Leu Leu Pro Arg Lys Gly Gly Arg Val Arg Phe Val Ala Lys
20 25 30
Lys Glu Leu Phe Tyr Val Pro Leu Leu Gly Trp Leu Leu Arg Leu Leu
35 40 45
Gly Ala Ile Phe Ile Asp Arg Glu Asn Gly Arg Leu Ala Arg Ala Ala
50 55 60
Leu Arg Glu Ala Val Arg Leu Leu Arg Asp Gly Gly Trp Leu Leu Ile
65 70 75 80
Phe Pro Glu Gly Thr Arg Ser Arg Pro Gly Lys Leu Leu Pro Phe Lys
85 90 95
Lys Gly Ala Ala Arg Leu Ala Leu Glu Ala Gly Val Pro Ile Val Pro
100 105 110
Val Ala Ile Arg Gly Thr
115
<210> 16
<211> 187
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> консервативная последовательность
<220>
<221> X
<222> (15)..(15)
<223> любая аминокислота
<220>
<221> X
<222> (18)..(18)
<223> любая аминокислота
<220>
<221> X
<222> (23)..(23)
<223> любая аминокислота
<220>
<221> X
<222> (25)..(26)
<223> любая аминокислота
<220>
<221> X
<222> (28)..(30)
<223> любая аминокислота
<220>
<221> X
<222> (32)..(33)
<223> любая аминокислота
<220>
<221> X
<222> (35)..(38)
<223> любая аминокислота
<220>
<221> X
<222> (41)..(41)
<223> любая аминокислота
<220>
<221> X
<222> (46)..(48)
<223> любая аминокислота
<220>
<221> X
<222> (53)..(53)
<223> любая аминокислота
<220>
<221> X
<222> (55)..(57)
<223> любая аминокислота
<220>
<221> X
<222> (61)..(61)
<223> любая аминокислота
<220>
<221> X
<222> (67)..(67)
<223> любая аминокислота
<220>
<221> X
<222> (72)..(72)
<223> любая аминокислота
<220>
<221> X
<222> (74)..(77)
<223> любая аминокислота
<220>
<221> X
<222> (79)..(79)
<223> любая аминокислота
<220>
<221> X
<222> (114)..(114)
<223> любая аминокислота
<220>
<221> X
<222> (127)..(128)
<223> любая аминокислота
<220>
<221> X
<222> (136)..(136)
<223> любая аминокислота
<220>
<221> X
<222> (139)..(142)
<223> любая аминокислота
<220>
<221> X
<222> (144)..(144)
<223> любая аминокислота
<220>
<221> X
<222> (150)..(150)
<223> любая аминокислота
<220>
<221> X
<222> (164)..(165)
<223> любая аминокислота
<220>
<221> X
<222> (167)..(172)
<223> любая аминокислота
<400> 16
Ala Val Phe Asp Lys Asp Gly Thr Leu Thr Glu Asp Asp Thr Xaa Phe
1 5 10 15
Leu Xaa Tyr Leu Leu Lys Xaa Leu Xaa Xaa Leu Xaa Xaa Xaa Leu Xaa
20 25 30
Xaa Asp Xaa Xaa Xaa Xaa Gly Ser Xaa Leu Thr Leu Ser Xaa Xaa Xaa
35 40 45
Asp Leu Leu Glu Xaa Leu Xaa Xaa Xaa Gly Gly Ile Xaa Val Ile Gly
50 55 60
Leu Ala Xaa Arg Tyr Leu Glu Xaa Leu Xaa Xaa Xaa Xaa Glu Xaa Ala
65 70 75 80
Lys Leu Phe Glu Gly Phe Ile Lys Pro Asp Ala Ala Glu Leu Leu Lys
85 90 95
Glu Leu His Glu Ala Gly Leu Arg Val Val Val Leu Thr Gly Asp Pro
100 105 110
Arg Xaa Ile Ala Lys Pro Val Ala Lys Glu Leu Gly Ile Asp Xaa Xaa
115 120 125
Asn Val Leu Ala Thr Glu Leu Xaa Asp Glu Xaa Xaa Xaa Xaa Val Xaa
130 135 140
Gly Arg Ile Thr Gly Xaa Leu Asp Lys Ala Arg Ala Val Glu Arg Leu
145 150 155 160
Val Val Leu Xaa Xaa Lys Xaa Xaa Xaa Xaa Xaa Xaa Val Val Ala Ile
165 170 175
Gly Asp Ser Ala Asn Asp Leu Pro Ala Leu Lys
180 185
<210> 17
<211> 190
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> консервативная последовательность
<400> 17
Ile Lys Ala Val Val Phe Asp Lys Asp Gly Thr Leu Thr Asp Gly Glu
1 5 10 15
Pro Pro Ile Ala Glu Ala Ile Val Glu Ala Ala Ala Glu Leu Gly Leu
20 25 30
Pro Leu Leu Leu Pro Leu Glu Glu Val Glu Lys Leu Leu Gly Arg Gly
35 40 45
Val Glu Gly Ile Glu Arg Ile Leu Leu Glu Gly Gly Leu Thr Ala Glu
50 55 60
Leu Leu Leu Glu Leu Glu Gly Glu Leu Ala Ala Gly Lys Thr Ala Val
65 70 75 80
Leu Val Ala Leu Asp Gly Glu Val Leu Gly Leu Ile Ala Leu Ala Asp
85 90 95
Lys Leu Tyr Pro Gly Ala Arg Glu Ala Leu Lys Ala Leu Lys Glu Arg
100 105 110
Gly Ile Lys Val Ala Ile Leu Thr Asn Gly Asp Arg Ala Asn Ala Glu
115 120 125
Ala Val Leu Glu Ala Leu Gly Leu Ala Asp Leu Phe Asp Val Ile Val
130 135 140
Asp Ser Asp Asp Val Gly Pro Val Lys Pro Lys Pro Glu Ile Phe Leu
145 150 155 160
Lys Ala Leu Glu Arg Leu Gly Val Lys Pro Glu Glu Val Leu Met Val
165 170 175
Gly Asp Gly Val Asn Asp Ala Pro Ala Leu Ala Ala Ala Gly
180 185 190
<210> 18
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> консервативная последовательность
<400> 18
Gly Asp Leu Val Ile Cys Pro Glu Gly Thr Thr Cys Arg Glu Pro
1 5 10 15
<210> 19
<211> 6
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> консервативная последовательность
<220>
<221> X
<222> (2)..(2)
<223> любая аминокислота
<220>
<221> X
<222> (4)..(4)
<223> любая аминокислота
<220>
<221> X
<222> (5)..(5)
<223> треонин (T) или валин (V)
<220>
<221> X
<222> (6)..(6)
<223> лейцин (L) или валин (V)
<400> 19
Asp Xaa Asp Xaa Xaa Xaa
1 5
<210> 20
<211> 8
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> консервативная последовательность
<220>
<221> X
<222> (2)..(2)
<223> 17-20 аминокислот; где аминокислоты могут представлять собой
любые аминокислоты
<220>
<221> X
<222> (3)..(3)
<223> глицин (G) или серин (S)
<220>
<221> X
<222> (4)..(4)
<223> аспарагиновая кислота (D) или серин (S)
<220>
<221> X
<222> (5)..(7)
<223> любая аминокислота
<220>
<221> X
<222> (8)..(8)
<223> аспарагиновая кислота (D) или аспарагин (N)
<400> 20
Lys Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5
<210> 21
<211> 356
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 21
Met Asp Trp Glu Ile Arg Gly Ser Ser Leu Gly Gln Lys Leu Leu Glu
1 5 10 15
Phe Asp Ser Glu Gln Glu Arg Gln Thr Arg Phe Arg Ala Tyr Asp Ser
20 25 30
Glu Glu Ala Ala Ala His Thr Tyr Asp Leu Ala Ala Leu Lys Tyr Trp
35 40 45
Gly Pro Asp Thr Ile Leu Asn Phe Pro Ala Glu Thr Tyr Thr Lys Glu
50 55 60
Leu Glu Glu Met Gln Arg Val Thr Lys Glu Glu Tyr Leu Ala Ser Leu
65 70 75 80
Arg Arg Gln Ser Ser Gly Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly
85 90 95
Val Ala Arg His His His Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg
100 105 110
Val Phe Gly Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr Asn Thr Gln Glu
115 120 125
Glu Ala Ala Ala Ala Tyr Asp Met Ala Ala Ile Glu Tyr Arg Gly Ala
130 135 140
Asn Ala Val Thr Asn Phe Asp Ile Ser Asn Tyr Ile Asp Arg Leu Lys
145 150 155 160
Lys Lys Gly Val Phe Pro Phe Pro Val Asn Gln Ala Asn His Gln Glu
165 170 175
Gly Ile Leu Val Glu Ala Lys Gln Glu Val Glu Thr Arg Glu Ala Lys
180 185 190
Glu Glu Pro Arg Glu Glu Val Lys Gln Gln Tyr Val Glu Glu Pro Pro
195 200 205
Gln Glu Glu Glu Glu Lys Glu Glu Glu Lys Ala Glu Gln Gln Glu Ala
210 215 220
Glu Ile Val Gly Tyr Ser Glu Glu Ala Ala Val Val Asn Cys Cys Ile
225 230 235 240
Asp Ser Ser Thr Ile Met Glu Met Asp Arg Cys Gly Asp Asn Asn Glu
245 250 255
Leu Ala Trp Asn Phe Cys Met Met Asp Thr Gly Phe Ser Pro Phe Leu
260 265 270
Thr Asp Gln Asn Leu Ala Asn Glu Asn Pro Ile Glu Tyr Pro Glu Leu
275 280 285
Phe Asn Glu Leu Ala Phe Glu Asp Asn Ile Asp Phe Met Phe Asp Asp
290 295 300
Gly Lys His Glu Cys Leu Asn Leu Glu Asn Leu Asp Cys Cys Val Val
305 310 315 320
Gly Arg Glu Ser Pro Pro Ser Ser Ser Ser Pro Leu Ser Cys Leu Ser
325 330 335
Thr Asp Ser Ala Ser Ser Thr Thr Thr Thr Thr Thr Ser Val Ser Cys
340 345 350
Asn Tyr Leu Val
355
<210> 22
<211> 430
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 22
Met Lys Lys Arg Leu Thr Thr Ser Thr Cys Ser Ser Ser Pro Ser Ser
1 5 10 15
Ser Val Ser Ser Ser Thr Thr Thr Ser Ser Pro Ile Gln Ser Glu Ala
20 25 30
Pro Arg Pro Lys Arg Ala Lys Arg Ala Lys Lys Ser Ser Pro Ser Gly
35 40 45
Asp Lys Ser His Asn Pro Thr Ser Pro Ala Ser Thr Arg Arg Ser Ser
50 55 60
Ile Tyr Arg Gly Val Thr Arg His Arg Trp Thr Gly Arg Phe Glu Ala
65 70 75 80
His Leu Trp Asp Lys Ser Ser Trp Asn Ser Ile Gln Asn Lys Lys Gly
85 90 95
Lys Gln Val Tyr Leu Gly Ala Tyr Asp Ser Glu Glu Ala Ala Ala His
100 105 110
Thr Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Pro Asp Thr Ile Leu
115 120 125
Asn Phe Pro Ala Glu Thr Tyr Thr Lys Glu Leu Glu Glu Met Gln Arg
130 135 140
Val Thr Lys Glu Glu Tyr Leu Ala Ser Leu Arg Arg Gln Ser Ser Gly
145 150 155 160
Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly Val Ala Arg His His His
165 170 175
Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg Val Phe Gly Asn Lys Tyr
180 185 190
Leu Tyr Leu Gly Thr Tyr Asn Thr Gln Glu Glu Ala Ala Ala Ala Tyr
195 200 205
Asp Met Ala Ala Ile Glu Tyr Arg Gly Ala Asn Ala Val Thr Asn Phe
210 215 220
Asp Ile Ser Asn Tyr Ile Asp Arg Leu Lys Lys Lys Gly Val Phe Pro
225 230 235 240
Phe Pro Val Asn Gln Ala Asn His Gln Glu Gly Ile Leu Val Glu Ala
245 250 255
Lys Gln Glu Val Glu Thr Arg Glu Ala Lys Glu Glu Pro Arg Glu Glu
260 265 270
Val Lys Gln Gln Tyr Val Glu Glu Pro Pro Gln Glu Glu Glu Glu Lys
275 280 285
Glu Glu Glu Lys Ala Glu Gln Gln Glu Ala Glu Ile Val Gly Tyr Ser
290 295 300
Glu Glu Ala Ala Val Val Asn Cys Cys Ile Asp Ser Ser Thr Ile Met
305 310 315 320
Glu Met Asp Arg Cys Gly Asp Asn Asn Glu Leu Ala Trp Asn Phe Cys
325 330 335
Met Met Asp Thr Gly Phe Ser Pro Phe Leu Thr Asp Gln Asn Leu Ala
340 345 350
Asn Glu Asn Pro Ile Glu Tyr Pro Glu Leu Phe Asn Glu Leu Ala Phe
355 360 365
Glu Asp Asn Ile Asp Phe Met Phe Asp Asp Gly Lys His Glu Cys Leu
370 375 380
Asn Leu Glu Asn Leu Asp Cys Cys Val Val Gly Arg Glu Ser Pro Pro
385 390 395 400
Ser Ser Ser Ser Pro Leu Ser Cys Leu Ser Thr Asp Ser Ala Ser Ser
405 410 415
Thr Thr Thr Thr Thr Thr Ser Val Ser Cys Asn Tyr Leu Val
420 425 430
<210> 23
<211> 430
<212> БЕЛОК
<213> Arabidopsis lyrata
<400> 23
Met Lys Arg Arg Leu Thr Thr Ser Thr Ser Ser Ser Ser Pro Ser Ser
1 5 10 15
Ser Val Ser Ser Ser Thr Thr Thr Ser Ser Pro Ile Gln Ser Glu Ala
20 25 30
Pro Arg Pro Lys Arg Ala Lys Arg Ala Lys Lys Ser Ser Pro Ser Gly
35 40 45
Asp Lys Ser His Asn Pro Thr Ser Pro Ala Ser Thr Arg Arg Ser Ser
50 55 60
Ile Tyr Arg Gly Val Thr Arg His Arg Trp Thr Gly Arg Phe Glu Ala
65 70 75 80
His Leu Trp Asp Lys Ser Ser Trp Asn Ser Ile Gln Asn Lys Lys Gly
85 90 95
Lys Gln Gly Ala Tyr Asp Ser Glu Glu Ala Ala Ala His Thr Tyr Asp
100 105 110
Leu Ala Ala Leu Lys Tyr Trp Gly Pro Asp Thr Ile Leu Asn Phe Pro
115 120 125
Ala Glu Thr Tyr Thr Lys Glu Leu Glu Glu Met Gln Arg Val Thr Lys
130 135 140
Glu Glu Tyr Leu Ala Ser Leu Arg Arg Gln Ser Ser Gly Phe Ser Arg
145 150 155 160
Gly Val Ser Lys Tyr Arg Gly Val Ala Arg His His His Asn Gly Arg
165 170 175
Trp Glu Ala Arg Ile Gly Arg Val Phe Gly Asn Lys Tyr Leu Tyr Leu
180 185 190
Gly Thr Tyr Asn Thr Gln Glu Glu Ala Ala Ala Ala Tyr Asp Met Ala
195 200 205
Ala Ile Glu Tyr Arg Gly Ala Asn Ala Val Thr Asn Phe Asp Ile Ser
210 215 220
Asn Tyr Ile Asp Arg Leu Lys Lys Lys Gly Val Phe Pro Phe Pro Val
225 230 235 240
Asn Gln Pro Asn His Gln Glu Ala Ile Leu Val Glu Ala Lys Gln Glu
245 250 255
Ile Glu Thr Arg Glu Ala Lys Glu Glu Pro Arg Glu Glu Val Lys Gln
260 265 270
Gln Tyr Val Glu Glu Pro Pro Gln Glu Glu Lys Glu Glu Glu Lys Ala
275 280 285
Glu Gln Gln Glu Ala Glu Phe Val Gly Tyr Lys Asp Glu Gly Ala Val
290 295 300
Val Asn Cys Cys Ile Asp Ser Ser Ala Ile Met Glu Met Asn Arg Cys
305 310 315 320
Gly Asp Asn Asn Glu Leu Ala Trp Asn Phe Cys Met Met Asp Ser Gly
325 330 335
Phe Ala Pro Phe Leu Thr Asp Gln Asn Leu Ser Asn Glu Asn Pro Ile
340 345 350
Glu Tyr Pro Glu Leu Phe Asn Glu Leu Ala Phe Glu Asp Asn Ile Asp
355 360 365
Phe Met Phe Asp Glu Ala Lys Asn Asp Cys Leu Ser Leu Glu Asn Leu
370 375 380
Asp Cys Cys Val Val Gly Arg Glu Ser Pro Thr Ser Ser Ser Ser Pro
385 390 395 400
Leu Ser Cys Phe Ser Thr Asp Ser Ala Ser Ser Thr Thr Thr Thr Thr
405 410 415
Ser Val Ser Cys Asn Tyr Leu Gly Leu Phe Val Gly Ser Glu
420 425 430
<210> 24
<211> 413
<212> БЕЛОК
<213> Brassica napus
<400> 24
Met Lys Arg Pro Leu Thr Thr Ser Pro Ser Ser Ser Ser Ser Thr Ser
1 5 10 15
Ser Ser Ala Cys Ile Leu Pro Thr Gln Ser Glu Thr Pro Arg Pro Lys
20 25 30
Arg Ala Lys Arg Ala Lys Lys Ser Ser Leu Arg Ser Asp Val Lys Pro
35 40 45
Gln Asn Pro Thr Ser Pro Ala Ser Thr Arg Arg Ser Ser Ile Tyr Arg
50 55 60
Gly Val Thr Arg His Arg Trp Thr Gly Arg Tyr Glu Ala His Leu Trp
65 70 75 80
Asp Lys Ser Ser Trp Asn Ser Ile Gln Asn Lys Lys Gly Lys Gln Val
85 90 95
Tyr Leu Gly Ala Tyr Asp Ser Glu Glu Ala Ala Ala His Thr Tyr Asp
100 105 110
Leu Ala Ala Leu Lys Tyr Trp Gly Pro Asn Thr Ile Leu Asn Phe Pro
115 120 125
Val Glu Thr Tyr Thr Lys Glu Leu Glu Glu Met Gln Arg Cys Thr Lys
130 135 140
Glu Glu Tyr Leu Ala Ser Leu Arg Arg Gln Ser Ser Gly Phe Ser Arg
145 150 155 160
Gly Val Ser Lys Tyr Arg Gly Val Ala Arg His His His Asn Gly Arg
165 170 175
Trp Glu Ala Arg Ile Gly Arg Val Phe Gly Asn Lys Tyr Leu Tyr Leu
180 185 190
Gly Thr Tyr Asn Thr Gln Glu Glu Ala Ala Ala Ala Tyr Asp Met Ala
195 200 205
Ala Ile Glu Tyr Arg Gly Ala Asn Ala Val Thr Asn Phe Asp Ile Gly
210 215 220
Asn Tyr Ile Asp Arg Leu Lys Lys Lys Gly Val Phe Pro Phe Pro Val
225 230 235 240
Ser Gln Ala Asn His Gln Glu Ala Val Leu Ala Glu Thr Lys Gln Glu
245 250 255
Val Glu Ala Lys Glu Glu Pro Thr Glu Glu Val Lys Gln Cys Val Glu
260 265 270
Lys Glu Glu Ala Lys Glu Glu Lys Thr Glu Lys Lys Gln Gln Gln Glu
275 280 285
Val Glu Glu Ala Val Ile Thr Cys Cys Ile Asp Ser Ser Glu Ser Asn
290 295 300
Glu Leu Ala Trp Asp Phe Cys Met Met Asp Ser Gly Phe Ala Pro Phe
305 310 315 320
Leu Thr Asp Ser Asn Leu Ser Ser Glu Asn Pro Ile Glu Tyr Pro Glu
325 330 335
Leu Phe Asn Glu Met Gly Phe Glu Asp Asn Ile Asp Phe Met Phe Glu
340 345 350
Glu Gly Lys Gln Asp Cys Leu Ser Leu Glu Asn Leu Asp Cys Cys Asp
355 360 365
Gly Val Val Val Val Gly Arg Glu Ser Pro Thr Ser Leu Ser Ser Ser
370 375 380
Pro Leu Ser Cys Leu Ser Thr Asp Ser Ala Ser Ser Thr Thr Thr Thr
385 390 395 400
Ala Thr Thr Val Thr Ser Val Ser Trp Asn Tyr Ser Val
405 410
<210> 25
<211> 415
<212> БЕЛОК
<213> Brassica napus
<400> 25
Met Lys Arg Pro Leu Thr Thr Ser Pro Ser Thr Ser Ser Ser Thr Ser
1 5 10 15
Ser Ser Ala Cys Ile Leu Pro Thr Gln Pro Glu Thr Pro Arg Pro Lys
20 25 30
Arg Ala Lys Arg Ala Lys Lys Ser Ser Ile Pro Thr Asp Val Lys Pro
35 40 45
Gln Asn Pro Thr Ser Pro Ala Ser Thr Arg Arg Ser Ser Ile Tyr Arg
50 55 60
Gly Val Thr Arg His Arg Trp Thr Gly Arg Tyr Glu Ala His Leu Trp
65 70 75 80
Asp Lys Ser Ser Trp Asn Ser Ile Gln Asn Lys Lys Gly Lys Gln Val
85 90 95
Tyr Leu Gly Ala Tyr Asp Ser Glu Glu Ala Ala Ala His Thr Tyr Asp
100 105 110
Leu Ala Ala Leu Lys Tyr Trp Gly Pro Asp Thr Ile Leu Asn Phe Pro
115 120 125
Ala Glu Thr Tyr Thr Lys Glu Leu Glu Glu Met Gln Arg Cys Thr Lys
130 135 140
Glu Glu Tyr Leu Ala Ser Leu Arg Arg Gln Ser Ser Gly Phe Ser Arg
145 150 155 160
Gly Val Ser Lys Tyr Arg Gly Val Ala Arg His His His Asn Gly Arg
165 170 175
Trp Glu Ala Arg Ile Gly Arg Val Phe Gly Asn Lys Tyr Leu Tyr Leu
180 185 190
Gly Thr Tyr Asn Thr Gln Glu Glu Ala Ala Ala Ala Tyr Asp Met Ala
195 200 205
Ala Ile Glu Tyr Arg Gly Ala Asn Ala Val Thr Asn Phe Asp Ile Ser
210 215 220
Asn Tyr Ile Asp Arg Leu Lys Lys Lys Gly Val Phe Pro Phe Pro Val
225 230 235 240
Ser Gln Ala Asn His Gln Glu Ala Val Leu Ala Glu Ala Lys Gln Glu
245 250 255
Val Glu Ala Lys Glu Glu Pro Thr Glu Glu Val Lys Gln Cys Val Glu
260 265 270
Lys Glu Glu Pro Gln Glu Ala Lys Glu Glu Lys Thr Glu Lys Lys Gln
275 280 285
Gln Gln Gln Glu Val Glu Glu Ala Val Val Thr Cys Cys Ile Asp Ser
290 295 300
Ser Glu Ser Asn Glu Leu Ala Trp Asp Phe Cys Met Met Asp Ser Gly
305 310 315 320
Phe Ala Pro Phe Leu Thr Asp Ser Asn Leu Ser Ser Glu Asn Pro Ile
325 330 335
Glu Tyr Pro Glu Leu Phe Asn Glu Met Gly Phe Glu Asp Asn Ile Asp
340 345 350
Phe Met Phe Glu Glu Gly Lys Gln Asp Cys Leu Ser Leu Glu Asn Leu
355 360 365
Asp Cys Cys Asp Gly Val Val Val Val Gly Arg Glu Ser Pro Thr Ser
370 375 380
Leu Ser Ser Ser Pro Leu Ser Cys Leu Ser Thr Asp Ser Ala Ser Ser
385 390 395 400
Thr Thr Thr Thr Thr Ile Thr Ser Val Ser Cys Asn Tyr Ser Val
405 410 415
<210> 26
<211> 285
<212> БЕЛОК
<213> Glycine max
<400> 26
Met Lys Arg Ser Pro Ala Ser Ser Cys Ser Ser Ser Thr Ser Ser Val
1 5 10 15
Gly Phe Glu Val His His Pro Ile Glu Lys Arg Arg Pro Lys His Pro
20 25 30
Arg Arg Asn Asn Leu Lys Ser Gln Lys Cys Lys Gln Asn Gln Thr Thr
35 40 45
Thr Gly Gly Arg Arg Ser Ser Ile Tyr Arg Gly Val Thr Arg His Arg
50 55 60
Trp Thr Gly Arg Phe Glu Ala His Leu Trp Asp Lys Ser Ser Trp Asn
65 70 75 80
Asn Ile Gln Ser Lys Lys Gly Lys Gln Val Tyr Leu Gly Ala Tyr Asp
85 90 95
Thr Glu Glu Ser Ala Ala Arg Thr Tyr Asp Leu Ala Ala Leu Lys Tyr
100 105 110
Trp Gly Lys Asp Ala Thr Leu Asn Phe Pro Ile Glu Thr Tyr Thr Lys
115 120 125
Asp Leu Glu Glu Met Asp Lys Val Ser Arg Glu Glu Tyr Leu Ala Ser
130 135 140
Leu Arg Arg Gln Ser Ser Gly Phe Ser Arg Gly Ile Ser Lys Tyr Arg
145 150 155 160
Gly Val Ala Arg His His His Asn Gly Arg Trp Glu Ala Arg Ile Gly
165 170 175
Arg Val Cys Gly Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr Lys Thr Gln
180 185 190
Glu Glu Ala Ala Val Ala Tyr Asp Met Ala Ala Ile Glu Tyr Arg Gly
195 200 205
Val Asn Ala Val Thr Asn Phe Asp Ile Ser Asn Tyr Met Asp Lys Ile
210 215 220
Lys Lys Lys Asn Asp Gln Thr Leu Gln Gln Gln Gln Thr Glu Val Gln
225 230 235 240
Thr Glu Thr Val Pro Asn Ser Ser Asp Ser Glu Glu Ala Glu Val Glu
245 250 255
Gln Gln His Thr Thr Thr Ile Thr Thr Pro Pro Pro Ser Glu Asn Leu
260 265 270
His Met Leu Pro Gln Glu His Gln Val Gly Gly Trp Val
275 280 285
<210> 27
<211> 417
<212> БЕЛОК
<213> Jatropha curcas
<400> 27
Met Lys Arg Ser Ser Ala Ser Ser Cys Ser Ser Ser Ser Ser Ser Ser
1 5 10 15
Ser Ser Pro Ser Ser Ser Ser Ser Ser Ala Cys Ser Ala Ser Ser Ser
20 25 30
Cys Leu Asp Ser Val Ser Pro Pro Asn His His Gln Leu Arg Ser Glu
35 40 45
Lys Ser Lys Ser Lys Arg Ile Arg Lys Ile Gln Thr Lys Gln Asp Lys
50 55 60
Cys Gln Thr Thr Ala Thr Thr Thr Ser Pro Ser Gly Gly Gly Arg Arg
65 70 75 80
Ser Ser Ile Tyr Arg Gly Val Thr Arg His Arg Trp Thr Gly Arg Phe
85 90 95
Glu Ala His Leu Trp Asp Lys Ser Ser Trp Asn Asn Ile Gln Asn Lys
100 105 110
Lys Gly Arg Gln Val Tyr Leu Gly Ala Tyr Asp Asn Glu Glu Ala Ala
115 120 125
Ala His Thr Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Gln Asp Thr
130 135 140
Thr Leu Asn Phe Pro Ile Glu Thr Tyr Ser Lys Glu Leu Glu Glu Met
145 150 155 160
Gln Lys Met Ser Lys Glu Glu Tyr Leu Ala Ser Leu Arg Arg Arg Ser
165 170 175
Ser Gly Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly Val Ala Arg His
180 185 190
His His Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg Val Phe Gly Asn
195 200 205
Lys Tyr Leu Tyr Leu Gly Thr Tyr Asn Thr Gln Glu Glu Ala Ala Ala
210 215 220
Ala Tyr Asp Met Ala Ala Ile Glu Tyr Arg Gly Ala Asn Ala Val Thr
225 230 235 240
Asn Phe Asp Val Ser His Tyr Ile Asp Arg Leu Lys Lys Lys Gly Ile
245 250 255
Pro Leu Asp Lys Ile Leu Pro Glu Thr Leu Ser Lys Gly Ser Lys Glu
260 265 270
Ser Glu Glu Ile Glu Arg Thr Ser Pro Leu Pro Leu Pro Ser Pro Pro
275 280 285
Ser Pro Ser Ile Thr Pro Leu His Glu Glu Ile Val Ser Pro Gln Leu
290 295 300
Leu Glu Thr Glu Cys Pro Gln His Pro Pro Cys Met Asp Thr Cys Thr
305 310 315 320
Met Ile Val Met Asp Pro Ile Glu Glu His Glu Leu Thr Trp Ser Phe
325 330 335
Cys Leu Asp Ser Gly Leu Val Pro Leu Pro Val Pro Asp Leu Pro Leu
340 345 350
Ala Asn Gly Cys Glu Leu Pro Asp Leu Leu Asp Asp Thr Gly Phe Glu
355 360 365
Asp Asn Ile Asp Leu Ile Phe Asp Ala Cys Cys Phe Gly Asn Asp Ala
370 375 380
Asn Pro Ala Asp Glu Asn Gly Lys Glu Arg Leu Ser Ser Ala Ser Thr
385 390 395 400
Ser Pro Ser Cys Ser Thr Thr Leu Thr Ser Val Ser Cys Asn Tyr Ser
405 410 415
Val
<210> 28
<211> 443
<212> БЕЛОК
<213> Ricinus communis
<400> 28
Met Lys Arg Ser Pro Thr Ser Pro Cys Ser Ser Ser Ser Ser Ser Ser
1 5 10 15
Tyr Ser Ser Ser Ser Ala Ser Ser Ser Cys Val Gly Pro Asp Asp Thr
20 25 30
Pro Val Ala Pro Gly Ser His His His His Asp His His Gln Leu Arg
35 40 45
Ser Gln Lys Ser Ser Lys Arg Ile Arg Lys Val Lys Lys Lys Gln Gln
50 55 60
Asn His Asn Ile Asp Gln Asn Asn Thr Asn Thr Thr Ile Thr Ala Pro
65 70 75 80
Thr Ser Ala Arg Arg Ser Ser Ile Tyr Arg Gly Val Thr Arg His Arg
85 90 95
Trp Thr Gly Arg Phe Glu Ala His Leu Trp Asp Lys Ser Ser Trp Asn
100 105 110
Asn Ile Gln Asn Lys Lys Gly Arg Gln Gly Ala Tyr Asp Asn Glu Glu
115 120 125
Ala Ala Ala His Thr Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Pro
130 135 140
Glu Thr Thr Leu Asn Phe Pro Ile Glu Thr Tyr Pro Lys Glu Leu Glu
145 150 155 160
Glu Met Gln Lys Met Ser Lys Glu Glu Tyr Leu Ala Ser Leu Arg Arg
165 170 175
Gln Ser Ser Gly Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly Val Ala
180 185 190
Arg His His His Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg Val Phe
195 200 205
Gly Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr Asn Thr Gln Glu Glu Ala
210 215 220
Ala Ala Ala Tyr Asp Met Ala Ala Ile Glu Tyr Arg Gly Ala Asn Ala
225 230 235 240
Val Thr Asn Phe Asp Ile Ser Asn Tyr Ile Asp Arg Leu Lys Lys Lys
245 250 255
Gly Ile Leu Leu Asp Gln Ile Leu Pro Asp Gln Pro Leu Arg Lys Cys
260 265 270
Ser Ser Glu Ser Glu Glu Ala Glu Ala Glu Ala Glu Val Glu Arg Leu
275 280 285
Pro Ser Leu Pro Ser Ser Ile Leu Pro Gln Glu Gln Asp Thr Ile Ser
290 295 300
Pro Gln Leu Gln Cys Thr Gln Leu Leu Pro Ser Met Asp Ser Cys Thr
305 310 315 320
Met Ile Asn Met Asp Pro Ile Glu Asp Asn Glu Leu Thr Trp Ser Phe
325 330 335
Cys Leu Asp Ser Gly Leu Thr Leu Phe Ser Val Pro Glu Leu Pro Leu
340 345 350
Glu Asn Ala Cys Glu Leu Pro Asp Leu Phe Asp Asp Thr Gly Phe Glu
355 360 365
Asp Asn Ile Asp Leu Ile Phe Asp Gly Cys Cys Phe Gly Asn Asp Asp
370 375 380
Asp Gly Gly Gly Gly Ala Asn His Gln Glu Phe Met Val Glu Ser Arg
385 390 395 400
Gly Cys Arg Val Gly Glu Val Gly Ile Ser Gly Ser Met Glu Glu Glu
405 410 415
Asn Gly Lys Glu Met Cys Cys Ser Ser Ser Ser Pro Ser Cys Ser Thr
420 425 430
Thr Thr Ser Val Ser Cys Cys Asn Tyr Ser Val
435 440
<210> 29
<211> 402
<212> БЕЛОК
<213> Populus trichocarpa
<400> 29
Met Lys Arg Ser Ser Ser Cys Ser Ser Ser Ser Ser Ser Ser Ser Ser
1 5 10 15
Ser Cys Val Ala Ser Glu Ser Ile His Lys Pro Lys Ala Lys Arg Ile
20 25 30
Arg Lys Asn Gln Lys Ser Asn Gln Gly Lys Ser Gln Asn Ala Ala Ala
35 40 45
Ala Ala Ala Asn Asn Ser His Asn Ser Gly Lys Arg Ser Ser Ile Tyr
50 55 60
Arg Gly Val Thr Arg His Arg Trp Thr Gly Arg Phe Glu Ala His Leu
65 70 75 80
Trp Asp Lys Ser Ser Trp Asn Ser Ile Gln Asn Lys Lys Gly Lys Gln
85 90 95
Gly Ala Tyr Asp Asn Glu Glu Ala Ala Ala His Thr Tyr Asp Leu Ala
100 105 110
Ala Leu Lys Tyr Trp Gly Ser Glu Thr Thr Leu Asn Phe Pro Ile Glu
115 120 125
Thr Tyr Thr Lys Glu Ile Glu Glu Met Gln Lys Val Thr Lys Glu Glu
130 135 140
Tyr Leu Ala Ser Leu Arg Arg Gln Ser Ser Gly Phe Ser Arg Gly Val
145 150 155 160
Ser Lys Tyr Arg Gly Val Ala Arg His His His Asn Gly Arg Trp Glu
165 170 175
Ala Arg Ile Gly Arg Val Tyr Gly Asn Lys Tyr Leu Tyr Leu Gly Thr
180 185 190
Tyr Asn Thr Gln Glu Glu Ala Ala Ala Ala Tyr Asp Met Ala Ala Ile
195 200 205
Gln Tyr Arg Gly Ala Asn Ala Val Thr Asn Phe Asp Val Ser Asn Tyr
210 215 220
Ile Glu Arg Leu Arg Lys Lys Gly Ile Pro Ile Asp Arg Ile Leu Gln
225 230 235 240
Glu Gln Gln Leu Leu Asn Asn Ser Val Asp Ser Ser Val Glu Val Glu
245 250 255
Val Glu Gln Pro Thr Pro Pro Pro Gln Gln Gln Gln Glu Glu Gln Glu
260 265 270
Gln Lys Ile Val Ser Ser Ser Ser Gln Leu Gln Cys Ser Gln Leu Asn
275 280 285
Ser Ser Leu Asp Gly Thr Pro Pro Met Val Ile Met Asp Thr Ile Glu
290 295 300
Glu His Glu Leu Ala Trp Ser Phe Cys Met Asp Ser Gly Leu Ser Leu
305 310 315 320
Thr Met Pro Asp Leu Pro Leu Glu Asn Ser Cys Glu Leu Pro Asp Leu
325 330 335
Phe Asp His Thr Gly Phe Glu Asp Asn Ile Asp Leu Ile Phe Asp Ala
340 345 350
Cys Cys Tyr Gly Lys Glu Ala Asn Pro Ala Gly Tyr Thr Leu Glu Asp
355 360 365
Asn Ser Thr Gly Gly Val Glu Glu Asp Arg Leu Ser Ser Asp Ser Val
370 375 380
Ser Asn Ser Pro Thr Ser Ser Thr Thr Thr Ser Val Ser Cys Asn Tyr
385 390 395 400
Ser Val
<210> 30
<211> 409
<212> БЕЛОК
<213> Vitis vinifera
<400> 30
Met Val Lys Arg Ser Ser Pro Gly Ser Ser Ser Ser Pro Ser Ser Ser
1 5 10 15
Ser Thr Ser Ser Asp Ala Ala Ser Arg Pro Ala Pro Pro Ser Gly Gly
20 25 30
Lys Pro Lys Ser Arg Lys Lys Glu Ala Lys Lys Asn Ser Asn Gly Asn
35 40 45
Gly Ser Asn Ser Lys Asn Lys Arg Thr Ser Ile Tyr Arg Gly Val Thr
50 55 60
Lys His Arg Trp Thr Gly Arg Phe Glu Ala His Leu Trp Asp Lys Ser
65 70 75 80
Ser Trp Asn Asp Ile Ser Asn Lys Arg Gly Arg Gln Gly Ala Tyr Tyr
85 90 95
Asn Glu Glu Ala Ala Ala Arg Thr Tyr Asp Leu Ala Ala Leu Lys Tyr
100 105 110
Trp Gly Pro Thr Thr Pro Leu Asn Phe Pro Leu Glu Thr Tyr Gln Lys
115 120 125
Asp Ala Glu Glu Met Glu Lys Met Ser Lys Glu Glu Tyr Leu Ala Leu
130 135 140
Leu Arg Arg Gln Ser Asn Gly Phe Ser Arg Gly Val Ser Lys His His
145 150 155 160
His Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg Val Leu Gly Asn Lys
165 170 175
Tyr Leu Tyr Leu Gly Thr Tyr Ser Thr Gln Glu Glu Ala Ala Ala Ala
180 185 190
Tyr Asp Met Ala Ala Ile Glu Tyr Arg Gly Leu Asn Ala Val Thr Asn
195 200 205
Phe Asp Ile Ser Asn Tyr Val Lys Leu Gly Arg Val Glu Ala Gln Val
210 215 220
Gln Glu Leu Ala Gln Gln Leu Gln Pro Asn Thr Pro Ile Gly Pro Gln
225 230 235 240
Asn Glu Leu Gln Lys Glu Glu Glu Glu Gln Leu Gln Glu Pro Val Leu
245 250 255
Ser Ser Ser Gln His Leu Pro Ser Met Asp Ser Ser Ala Met Glu Ile
260 265 270
Met Asp Pro Ala Asp Asp Pro Asp Leu Pro Trp Asn Phe Cys Ala Tyr
275 280 285
Ser Thr Leu Leu Val Pro Asp Val Pro Leu Gly Lys Gly Gly Glu Leu
290 295 300
Ser Asp Leu Phe Tyr Glu Lys Gly Phe Glu Asp Asn Ile Asp Tyr Met
305 310 315 320
Phe Glu Gly Ala Ala Gly Asn Glu Glu Glu Ser Asn Ser Ala Glu Asn
325 330 335
Gly Val Lys Glu Asn Gly Phe Met His Glu Leu Glu Val Asp Gly Lys
340 345 350
Leu Gln Asn Val Val Gly Phe Phe Phe Leu Ser Phe Phe Phe Leu Pro
355 360 365
Lys Arg Ala Gly Ile Arg Lys Arg Gly Val Asp Ser Cys Met Gln Leu
370 375 380
Phe Leu Tyr Phe Val Phe Leu Phe Tyr Pro Phe Leu Pro Glu Val Ser
385 390 395 400
Lys Phe Leu Phe His Leu Ser Leu Asp
405
<210> 31
<211> 420
<212> БЕЛОК
<213> Brachypodium distachyon
<400> 31
Met Lys Arg Ser Pro Pro Gln Pro Ser Pro Ser Pro Ser Ser Ser Pro
1 5 10 15
Ala Ser Ser Ser Ser Ser Pro Ser Ser Ser Asp Ser Ser Ser Ser Ile
20 25 30
Ala Ile Pro Arg Lys Arg Ala Arg Thr Ala Ala Ala Ala Ala Gly Gly
35 40 45
Gly Lys Ala Arg Ala Ala Ala Ala Lys Arg Pro Lys Lys Asp Gly Lys
50 55 60
Asp Ser Gly Ser Ser Ser Asn Gly Gly Gly Gly Gly Gly Gly Lys Arg
65 70 75 80
Ser Ser Ile Tyr Arg Gly Val Thr Arg His Arg Trp Thr Gly Arg Phe
85 90 95
Glu Ala His Leu Trp Asp Lys Asn Cys Phe Thr Ser Leu Gln Asn Lys
100 105 110
Lys Lys Gly Arg Gln Val Tyr Leu Gly Ala Tyr Asp Thr Glu Glu Ala
115 120 125
Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Pro Glu
130 135 140
Thr Thr Leu Asn Phe Ser Ala Asp Asp Tyr Gly Lys Glu Arg Ser Glu
145 150 155 160
Met Glu Ala Val Ser Arg Glu Glu Tyr Leu Ala Ala Leu Arg Arg Arg
165 170 175
Ser Ser Gly Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly Val Ala Arg
180 185 190
His His His Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg Val Leu Gly
195 200 205
Asn Lys Tyr Leu Tyr Leu Gly Thr Phe Asp Thr Gln Glu Glu Ala Ala
210 215 220
Arg Ala Tyr Asp Leu Ala Ala Ile Gln Tyr Arg Gly Ala Asn Ala Val
225 230 235 240
Thr Asn Phe Asp Ile Ser Arg Tyr Leu Asp Gln Pro Gln Leu Leu Glu
245 250 255
Gln Leu Gln Gln Gln Gln Gly Pro Gln Val Val Ala Ala Leu Gln Glu
260 265 270
Glu Ala Gln Arg Asp His Gln Ser Asp Asn Ala Val Gln Glu Leu Asn
275 280 285
Ser Gly Glu Ala Gln Thr Pro Gly Gly Ile Asp Glu Pro Ile Ala Ile
290 295 300
Gly Asp Ser Thr Glu Asp Ile Asn Thr Ser Leu Thr Val Asp Asp Ile
305 310 315 320
Ile Glu Glu Ser Leu Trp Ser Pro Tyr Glu Phe Asp Ile Met Ala Gly
325 330 335
Val Asn Val Ser Asn Ser Met Asn Leu Ser Glu Leu Phe Ser Asp Val
340 345 350
Ala Phe Glu Gly Asn Ile Gly Cys Leu Phe Glu Glu Cys Ser Gly Ile
355 360 365
Asp Asp Cys Ser Ser Arg His Gly Ala Gly Leu Ala Ala Phe Gly Leu
370 375 380
Phe Thr Glu Gly Asp Asp Lys Leu Lys Asp Val Ser Glu Met Glu Met
385 390 395 400
Glu Val Asn Pro Gln Ala Asn Asp Val Ser Cys Pro Pro Lys Met Ile
405 410 415
Thr Val Cys Asn
420
<210> 32
<211> 423
<212> БЕЛОК
<213> Hordeum vulgare
<400> 32
Met Lys Arg Ser Pro Pro Pro Gln Pro Ser Pro Ser Ser Ser Pro Ala
1 5 10 15
Cys Ser Pro Ser Pro Ser Ser Pro Ser Ser Ser Asp Ser Ser Ser Ile
20 25 30
Ala Ile Pro Arg Lys Arg Ala Arg Thr Gln Lys Ala Gly Ser Ala Lys
35 40 45
Ala Lys Ala Ala Pro Lys Arg Ala Lys Lys Asp Ser Gly Arg Ser Thr
50 55 60
Lys Asp Ser Asp Ala Ser Ala Asn Gly Ala Ala Ala Ser Gly Lys Arg
65 70 75 80
Ser Ser Ile Tyr Arg Gly Val Thr Arg His Arg Trp Thr Gly Arg Phe
85 90 95
Glu Ala His Leu Trp Asp Lys Asn Cys Phe Thr Ser Ile Gln Asn Lys
100 105 110
Lys Lys Gly Arg Gln Val Tyr Leu Gly Ala Tyr Asp Thr Glu Glu Ala
115 120 125
Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Pro Glu
130 135 140
Thr Thr Leu Asn Phe Thr Val Asp Glu Tyr Ala Lys Glu Arg Ser Glu
145 150 155 160
Met Glu Ala Val Ser Arg Glu Glu Tyr Leu Ala Ala Leu Arg Arg Arg
165 170 175
Ser Ser Gly Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly Val Ala Arg
180 185 190
His His His Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg Val Leu Gly
195 200 205
Asn Lys Tyr Leu Tyr Leu Gly Thr Phe Asp Thr Gln Glu Glu Ala Ala
210 215 220
Arg Ala Tyr Asp Leu Ala Ala Ile Glu Tyr Arg Gly Ala Asn Ala Val
225 230 235 240
Thr Asn Phe Asp Ile Ser Arg Tyr Leu Asp Gln Pro Gln Leu Leu Ala
245 250 255
Gln Leu Glu Gln Gly Pro Gln Val Val Pro Ala Leu Gln Glu Glu Leu
260 265 270
Gln His Asp His Gln Ser Asp Asn Ala Val Gln Glu Leu Asn Ser Gly
275 280 285
Glu Ala Gln Lys Pro Gly Ser Val Ser Glu Pro Ile Ala Val Asp Asp
290 295 300
Thr Asp Asn Thr Gly Asp Ile Gly Ala Pro Leu Val Phe Asp Ser Gly
305 310 315 320
Val Glu Glu Asn Leu Trp Ser Pro Cys Met Asp Tyr Asp Val Asp Pro
325 330 335
Ile Phe Gly Pro Asn Ile Ser Ser Ser Met Asn Leu Ser Glu Trp Phe
340 345 350
Asn Asp Pro Ala Phe Glu Ser Asn Ile Gly Tyr Met Phe Glu Gly Cys
355 360 365
Ser Asp Val Asp Asp Cys Ser Thr Arg His Gly Ala Gly Leu Ser Ala
370 375 380
Leu Gly Phe Leu Lys Glu Gly Asp Asp Lys Leu Lys Asp Gly Ser Asp
385 390 395 400
Met Glu Ala Glu Ile Thr Pro Gln Ala Asn Asp Val Ser Cys Pro Pro
405 410 415
Lys Met Ile Thr Val Cys Asn
420
<210> 33
<211> 443
<212> БЕЛОК
<213> Oryza sativa
<400> 33
Met Ala Lys Arg Ser Ser Pro Asp Pro Ala Ser Ser Ser Pro Ser Ala
1 5 10 15
Ser Ser Ser Pro Ser Ser Pro Ser Ser Ser Ser Ser Glu Asp Ser Ser
20 25 30
Ser Pro Met Ser Met Pro Cys Lys Arg Arg Ala Arg Pro Arg Thr Asp
35 40 45
Lys Ser Thr Gly Lys Ala Lys Arg Pro Lys Lys Glu Ser Lys Glu Val
50 55 60
Val Asp Pro Ser Ser Asn Gly Gly Gly Gly Gly Lys Arg Ser Ser Ile
65 70 75 80
Tyr Arg Gly Val Thr Arg His Arg Trp Thr Gly Arg Phe Glu Ala His
85 90 95
Leu Trp Asp Lys Asn Cys Ser Thr Ser Leu Gln Asn Lys Lys Lys Gly
100 105 110
Arg Gln Gly Ala Tyr Asp Ser Glu Glu Ala Ala Ala Arg Ala Tyr Asp
115 120 125
Leu Ala Ala Leu Lys Tyr Trp Gly Pro Glu Thr Val Leu Asn Phe Pro
130 135 140
Leu Glu Glu Tyr Glu Lys Glu Arg Ser Glu Met Glu Gly Val Ser Arg
145 150 155 160
Glu Glu Tyr Leu Ala Ser Leu Arg Arg Arg Ser Ser Gly Phe Ser Arg
165 170 175
Gly Val Ser Lys Tyr Arg Gly Val Ala Arg His His His Asn Gly Arg
180 185 190
Trp Glu Ala Arg Ile Gly Arg Val Leu Gly Asn Lys Tyr Leu Tyr Leu
195 200 205
Gly Thr Phe Asp Thr Gln Glu Glu Ala Ala Lys Ala Tyr Asp Leu Ala
210 215 220
Ala Ile Glu Tyr Arg Gly Ala Asn Ala Val Thr Asn Phe Asp Ile Ser
225 230 235 240
Cys Tyr Leu Asp Gln Pro Gln Leu Leu Ala Gln Leu Gln Gln Glu Pro
245 250 255
Gln Leu Leu Ala Gln Leu Gln Gln Glu Pro Gln Val Val Pro Ala Leu
260 265 270
His Glu Glu Pro Gln Asp Asp Asp Arg Ser Glu Asn Ala Val Gln Glu
275 280 285
Leu Ser Ser Ser Glu Ala Asn Thr Ser Ser Asp Asn Asn Glu Pro Leu
290 295 300
Ala Ala Asp Asp Ser Ala Glu Cys Met Asn Glu Pro Leu Pro Ile Val
305 310 315 320
Asp Gly Ile Glu Glu Ser Leu Trp Ser Pro Cys Leu Asp Tyr Glu Leu
325 330 335
Asp Thr Met Pro Gly Ala Tyr Phe Ser Asn Ser Met Asn Phe Ser Glu
340 345 350
Trp Phe Asn Asp Glu Ala Phe Glu Gly Gly Met Glu Tyr Leu Phe Glu
355 360 365
Gly Cys Ser Ser Ile Thr Glu Gly Gly Asn Ser Met Asp Asn Ser Gly
370 375 380
Val Thr Glu Tyr Asn Leu Phe Glu Glu Cys Asn Met Leu Glu Lys Asp
385 390 395 400
Ile Ser Asp Phe Leu Asp Lys Asp Ile Ser Asp Phe Leu Asp Lys Asp
405 410 415
Ile Ser Ile Ser Asp Gly Glu Arg Ile Ser Pro Gln Ala Asn Asn Ile
420 425 430
Ser Cys Pro Gln Lys Met Ile Ser Val Cys Asn
435 440
<210> 34
<211> 420
<212> БЕЛОК
<213> Sorghum bicolor
<400> 34
Met Asp Met Glu Arg Ser Gln Gln Gln Lys Ser Pro Thr Glu Ser Pro
1 5 10 15
Pro Pro Pro Ser Pro Ser Ser Ser Ser Ser Ser Val Ser Ala Asp Thr
20 25 30
Val Leu Pro Pro Pro Gly Lys Arg Arg Arg Ala Ala Thr Thr Ala Lys
35 40 45
Ala Lys Ala Gly Ala Lys Pro Lys Arg Ala Arg Lys Asp Ala Ala Ala
50 55 60
Ala Ala Asp Pro Pro Pro Pro Pro Ala Ala Ala Ala Ala Gly Lys Arg
65 70 75 80
Ser Ser Val Tyr Arg Gly Val Thr Arg His Arg Trp Thr Gly Arg Phe
85 90 95
Glu Ala His Leu Trp Asp Lys His Cys Leu Ala Ala Leu His Asn Lys
100 105 110
Lys Lys Gly Arg Gln Val Tyr Leu Gly Ala Tyr Asp Ser Glu Glu Ala
115 120 125
Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Pro Glu
130 135 140
Thr Leu Leu Asn Phe Pro Val Glu Asp Tyr Ser Ser Glu Met Pro Glu
145 150 155 160
Met Glu Gly Val Ser Arg Glu Glu Tyr Leu Ala Ser Leu Arg Arg Arg
165 170 175
Ser Ser Gly Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly Val Ala Arg
180 185 190
His His His Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg Val Phe Gly
195 200 205
Asn Lys Tyr Leu Tyr Leu Gly Thr Phe Asp Thr Gln Glu Glu Ala Ala
210 215 220
Lys Ala Tyr Asp Leu Ala Ala Ile Glu Tyr Arg Gly Val Asn Ala Val
225 230 235 240
Thr Asn Phe Asp Ile Ser Cys Tyr Leu Asp His Pro Leu Phe Leu Ala
245 250 255
Gln Leu Gln Gln Glu Pro Gln Val Val Pro Ala Leu Asn Gln Glu Ala
260 265 270
Gln Pro Asp Gln Ser Glu Thr Glu Thr Ile Ala Gln Glu Ser Val Ser
275 280 285
Ser Glu Ala Lys Thr Pro Asp Asp Asn Ala Glu Pro Asp Asp Asn Ala
290 295 300
Glu Pro Asp Asp Ile Ala Glu Pro Leu Ile Thr Val Asp Asp Ser Ile
305 310 315 320
Glu Glu Ser Leu Trp Ser Pro Cys Met Asp Tyr Glu Leu Asp Thr Met
325 330 335
Ser Arg Ser Asn Phe Gly Ser Ser Ile Asn Leu Ser Glu Trp Phe Asn
340 345 350
Asp Ala Asp Phe Asp Ser Asn Ile Gly Cys Leu Phe Asp Gly Cys Ser
355 360 365
Ala Val Asp Glu Gly Gly Lys Asp Gly Val Gly Leu Ala Asp Phe Ser
370 375 380
Leu Leu Glu Asp Phe Ser Leu Phe Glu Ala Gly Asp Gly Gln Leu Lys
385 390 395 400
Asp Val Leu Ser Asp Met Glu Glu Gly Ile Gln Pro Pro Thr Met Ile
405 410 415
Ser Val Cys Asn
420
<210> 35
<211> 395
<212> БЕЛОК
<213> Zea mays
<400> 35
Met Glu Arg Ser Gln Arg Gln Ser Pro Pro Pro Pro Ser Pro Ser Ser
1 5 10 15
Ser Ser Ser Ser Val Ser Ala Asp Thr Val Leu Val Pro Pro Gly Lys
20 25 30
Arg Arg Arg Ala Ala Thr Ala Lys Ala Gly Ala Glu Pro Asn Lys Arg
35 40 45
Ile Arg Lys Asp Pro Ala Ala Ala Ala Ala Gly Lys Arg Ser Ser Val
50 55 60
Tyr Arg Gly Val Thr Arg His Arg Trp Thr Gly Arg Phe Glu Ala His
65 70 75 80
Leu Trp Asp Lys His Cys Leu Ala Ala Leu His Asn Lys Lys Lys Gly
85 90 95
Arg Gln Val Tyr Leu Gly Ala Tyr Asp Ser Glu Glu Ala Ala Ala Arg
100 105 110
Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Pro Glu Thr Leu Leu
115 120 125
Asn Phe Pro Val Glu Asp Tyr Ser Ser Glu Met Pro Glu Met Glu Ala
130 135 140
Val Ser Arg Glu Glu Tyr Leu Ala Ser Leu Arg Arg Arg Ser Ser Gly
145 150 155 160
Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly Val Ala Arg His His His
165 170 175
Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg Val Phe Gly Asn Lys Tyr
180 185 190
Leu Tyr Leu Gly Thr Phe Asp Thr Gln Glu Glu Ala Ala Lys Ala Tyr
195 200 205
Asp Leu Ala Ala Ile Glu Tyr Arg Gly Val Asn Ala Val Thr Asn Phe
210 215 220
Asp Ile Ser Cys Tyr Leu Asp His Pro Leu Phe Leu Ala Gln Leu Gln
225 230 235 240
Gln Glu Pro Gln Val Val Pro Ala Leu Asn Gln Glu Pro Gln Pro Asp
245 250 255
Gln Ser Glu Thr Gly Thr Thr Glu Gln Glu Pro Glu Ser Ser Glu Ala
260 265 270
Lys Thr Pro Asp Gly Ser Ala Glu Pro Asp Glu Asn Ala Val Pro Asp
275 280 285
Asp Thr Ala Glu Pro Leu Ser Thr Val Asp Asp Ser Ile Glu Glu Gly
290 295 300
Leu Trp Ser Pro Cys Met Asp Tyr Glu Leu Asp Thr Met Ser Arg Pro
305 310 315 320
Asn Phe Gly Ser Ser Ile Asn Leu Ser Glu Trp Phe Ala Asp Ala Asp
325 330 335
Phe Asp Cys Asn Ile Gly Cys Leu Phe Asp Gly Cys Ser Ala Ala Asp
340 345 350
Glu Gly Ser Lys Asp Gly Val Gly Leu Ala Asp Phe Ser Leu Phe Glu
355 360 365
Ala Gly Asp Val Gln Leu Lys Asp Val Leu Ser Asp Met Glu Glu Gly
370 375 380
Ile Gln Pro Pro Ala Met Ile Ser Val Cys Asn
385 390 395
<210> 36
<211> 413
<212> БЕЛОК
<213> Brachypodium distachyon
<400> 36
Met Glu Ala Tyr Cys Ser Thr Leu Val Lys Asp Glu Leu Ile Asn Gly
1 5 10 15
Gly Gly Gly Gly Ser Ala Gly Gly Met Arg Tyr Cys Glu Ala Ala Pro
20 25 30
Arg Val Ser Pro Pro Val Ala Ile Lys Ser Val Lys Arg Arg Lys Arg
35 40 45
Glu Pro Pro Ala Val Ser Gly Met Thr Thr Val Ser Gly Gly Gly Lys
50 55 60
Asp Gly Asp Lys Ser Ala Gly Asn Ala Ala Ala Lys Arg Ser Ser Arg
65 70 75 80
Phe Arg Gly Val Ser Arg His Arg Trp Thr Gly Arg Phe Glu Ala His
85 90 95
Leu Trp Asp Lys Gly Thr Trp Asn Pro Thr Gln Lys Lys Lys Gly Lys
100 105 110
Gln Val Tyr Leu Gly Ala Tyr Asn Glu Glu Glu Ala Ala Ala Arg Ala
115 120 125
Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Pro Thr Thr Tyr Thr Asn
130 135 140
Phe Pro Val Val Asp Tyr Glu Lys Glu Leu Lys Val Met Gln Gly Val
145 150 155 160
Ser Arg Glu Glu Tyr Leu Ala Ser Ile Arg Arg Lys Ser Asn Gly Phe
165 170 175
Ser Arg Gly Val Ser Lys Tyr Arg Gly Val Ala Arg His His His Asn
180 185 190
Gly Arg Trp Glu Ala Arg Ile Gly Arg Val Phe Gly Asn Lys Tyr Leu
195 200 205
Tyr Leu Gly Thr Tyr Ser Thr Gln Glu Glu Ala Ala Arg Ala Tyr Asp
210 215 220
Ile Ala Ala Ile Glu Tyr Arg Gly Ile Asn Ala Val Thr Asn Phe Asp
225 230 235 240
Leu Ser Ser Tyr Ile Arg Trp Leu Lys Pro Asn Ser Thr Ile Asn Thr
245 250 255
Asn Thr Pro Ala Ala Glu Leu Ala Ile Leu Gly Gly Gly Gly Thr Pro
260 265 270
Ala Ala Leu Ile Thr Pro Pro Pro Thr Met His Val Pro Arg Leu Leu
275 280 285
Pro Pro Leu Val Lys Gly Arg Gly Ser Ser Ile Ala Asp Asp Val Ser
290 295 300
Ala Gly Ser Cys Val Phe Gly Gly Pro Ser Pro Ser Pro Ser Pro Thr
305 310 315 320
Thr Thr Ala Leu Ser Leu Leu Leu Arg Ser Ser Val Phe Gln Glu Leu
325 330 335
Val Ala Gln Gln Gln Pro Pro Ser Thr Val Asp Asp Asp Asp Asp Ile
340 345 350
Gly Gly His Ala Ala Val Ser Asp Ala Ala Gln Arg Ala Ala Glu Glu
355 360 365
Asn Glu Glu Ser Phe Gly Glu Val Leu Tyr Gly Ala Gly Glu Gly Glu
370 375 380
Ala Ala Thr Ala Phe Ser Cys Ser Met Tyr Glu Leu Gly Leu Asp Asp
385 390 395 400
Asn Phe Ala Arg Ile Glu Glu Ser Leu Trp Gly Cys Leu
405 410
<210> 37
<211> 423
<212> БЕЛОК
<213> Brachypodium sylvaticum
<400> 37
Met Glu Ala Tyr Cys Ser Ser Leu Val Lys Asp Glu Leu Ile Asn Gly
1 5 10 15
Gly Gly Gly Gly Ala Gly Gly Met Arg Tyr Cys Glu Ala Ala Pro Arg
20 25 30
Val Ser Pro Pro Val Ala Ile Lys Ser Val Lys Arg Arg Lys Arg Glu
35 40 45
Pro Pro Ala Val Ser Gly Met Thr Thr Val Ser Gly Gly Gly Gly Gly
50 55 60
Asn Gly Lys Asp Gly Asp Lys Ser Ala Gly Asn Ala Ala Ala Ala Lys
65 70 75 80
Arg Ser Ser Arg Phe Arg Gly Val Ser Arg His Arg Trp Thr Gly Arg
85 90 95
Phe Glu Ala His Leu Trp Asp Lys Gly Thr Trp Asn Pro Thr Gln Lys
100 105 110
Lys Lys Gly Lys Gln Val Tyr Leu Gly Ala Tyr Asn Glu Glu Glu Ala
115 120 125
Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Pro Thr
130 135 140
Thr Tyr Thr Asn Phe Pro Val Val Asp Tyr Glu Lys Glu Leu Lys Val
145 150 155 160
Met Gln Gly Val Ser Arg Glu Glu Tyr Leu Ala Ser Ile Arg Arg Lys
165 170 175
Ser Asn Gly Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly Val Ala Arg
180 185 190
His His His Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg Val Phe Gly
195 200 205
Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr Ser Thr Gln Glu Glu Ala Ala
210 215 220
Arg Ala Tyr Asp Ile Ala Ala Ile Glu Tyr Arg Gly Ile Asn Ala Val
225 230 235 240
Thr Asn Phe Asp Leu Ser Ser Tyr Ile Arg Trp Leu Lys Pro Asn Ser
245 250 255
Ala Ala Asn Thr Asn Thr Pro Pro Ala Ala Ala Ala Glu Leu Ala Ile
260 265 270
Leu Gly Gly Ala Pro Ala Ala Leu Ile Ser Pro Ala Pro Ala Pro Thr
275 280 285
Thr Met Arg Val Pro Arg Leu Leu Pro Pro Leu Val Arg Gly Arg Gly
290 295 300
Gly Ser Ile Pro Asp Asp Val Ser Ala Gly Gly Ser Cys Val Phe Gly
305 310 315 320
Ser Pro Ser Pro Ser Pro Ser Pro Thr Thr Thr Ser Ala Leu Ser Leu
325 330 335
Leu Leu Arg Ser Ser Val Phe Gln Glu Leu Val Ala Gln Gln Gln Pro
340 345 350
Pro Ser Ile Val Asp Asp Asp Asp Gly Val Gly Gly Gln Glu Ala Val
355 360 365
Ser Asp Ala Ala Glu Arg Ala Ala Glu Glu Asn Glu Glu Ser Phe Gly
370 375 380
Glu Val Leu Tyr Gly Ala Gly Glu Gly Glu Ala Ala Ala Ala Phe Ser
385 390 395 400
Cys Ser Met Tyr Glu Leu Gly Leu Asp Asp Ser Phe Ala Arg Ile Glu
405 410 415
Glu Ser Leu Trp Gly Cys Leu
420
<210> 38
<211> 399
<212> БЕЛОК
<213> Oryza sativa
<400> 38
Met Glu Thr Tyr Gly Leu Val Lys Asp Glu Leu Leu His Gly Ile Gly
1 5 10 15
Gly Gly Gln Gly Arg Leu Tyr Cys Glu Val Lys Pro Thr Ala Ala Pro
20 25 30
Ala Val Ile Thr Ala Ala Gly Gly Gly Ala Lys Ser Val Lys Arg Arg
35 40 45
Lys Arg Glu Pro Ser Ala Ala Ala Met Ser Ala Val Thr Val Ala Gly
50 55 60
Asn Gly Lys Glu Ala Gly Gly Ser Asn Ala Ala Asn Lys Arg Ser Ser
65 70 75 80
Arg Phe Arg Gly Val Ser Arg His Arg Trp Thr Gly Arg Phe Glu Ala
85 90 95
His Leu Trp Asp Lys Gly Thr Trp Asn Pro Thr Gln Lys Lys Lys Gly
100 105 110
Lys Gln Val Tyr Leu Gly Ala Tyr Asn Glu Glu Asp Ala Ala Ala Arg
115 120 125
Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Pro Thr Thr Tyr Thr
130 135 140
Asn Phe Pro Val Ala Asp Tyr Glu Lys Glu Leu Lys Leu Met Gln Gly
145 150 155 160
Val Ser Lys Glu Glu Tyr Leu Ala Ser Ile Arg Arg Lys Ser Asn Gly
165 170 175
Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly Val Ala Arg His His His
180 185 190
Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg Val Phe Gly Asn Lys Tyr
195 200 205
Leu Tyr Leu Gly Thr Tyr Ser Thr Gln Glu Glu Ala Ala Arg Ala Tyr
210 215 220
Asp Ile Ala Ala Ile Glu Tyr Arg Gly Ile Asn Ala Val Thr Asn Phe
225 230 235 240
Asp Leu Ser Thr Tyr Ile Arg Trp Leu Lys Pro Pro Ser Ser Ser Ser
245 250 255
Ala Ala Gly Thr Pro His His His Gly Gly Gly Met Val Val Gly Ala
260 265 270
Asp Arg Val Leu Ala Pro Ala Gln Ser Tyr Pro Ile Ser Ala Ala Ala
275 280 285
Asp Asp Asp Val Ala Gly Cys Trp Arg Pro Leu Pro Ser Pro Ser Ser
290 295 300
Ser Thr Thr Thr Ala Leu Ser Leu Leu Leu Arg Ser Ser Met Phe Gln
305 310 315 320
Glu Leu Val Ala Arg Gln Pro Val Val Glu Gly Asp Asp Gly Gln Leu
325 330 335
Ala Val Val Ser Gly Asp Asp Ala Asp Ala Asp Ala Asp Ser Asp Val
340 345 350
Lys Glu Pro Pro Pro Glu Ser Glu Tyr Gly Glu Val Phe Ala Ser Asp
355 360 365
Glu Ala Ala Ala Ala Ala Ala Tyr Gly Cys Ser Met Tyr Glu Leu Asp
370 375 380
Asp Ser Phe Ala Leu Ile Asp Asp Ser Val Trp Asn Cys Leu Ile
385 390 395
<210> 39
<211> 488
<212> БЕЛОК
<213> Sorghum bicolor
<400> 39
Met Glu Thr Tyr Ser Leu Gln Val Lys Asp Glu Leu His Gly Gly Gly
1 5 10 15
Ile Gly Ile Gly Gly Gly Gly Gln Gly Leu Tyr Cys Gly Ala Thr Pro
20 25 30
Arg Pro Ala Ala Pro Ala Ala Thr Gly Gly Gly Gly Gly Gly Gly Asp
35 40 45
Gly Ala Val Lys Ser Asn Lys Arg Ser Arg Lys Arg Glu Pro Pro Pro
50 55 60
Pro Pro Pro Ser Ser Leu Val Thr Met Ser Asn Gly Gly Lys Asp Glu
65 70 75 80
Ala Val Ala Gly Ser Gly Asp Lys Ser Ala Ser Ser Asn Ser Asn Ala
85 90 95
Ser Lys Arg Ser Ser Arg Phe Arg Gly Val Ser Arg His Arg Trp Thr
100 105 110
Gly Arg Phe Glu Ala His Leu Trp Asp Lys Gly Thr Trp Asn Pro Thr
115 120 125
Gln Lys Lys Lys Gly Lys Gln Val Tyr Leu Gly Ala Tyr Asn Glu Glu
130 135 140
Asp Ala Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly
145 150 155 160
Pro Thr Thr Tyr Thr Asn Phe Pro Val Val Asp Tyr Glu Arg Glu Leu
165 170 175
Lys Val Met Gln Asn Val Ser Lys Glu Glu Tyr Leu Ala Ser Ile Arg
180 185 190
Arg Lys Ser Asn Gly Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly Val
195 200 205
Ala Arg His His His Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg Val
210 215 220
Phe Gly Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr Ser Thr Gln Glu Glu
225 230 235 240
Ala Ala Arg Ala Tyr Asp Ile Ala Ala Ile Glu Tyr Arg Gly Ile Asn
245 250 255
Ala Val Thr Asn Phe Asp Leu Ser Thr Tyr Ile Arg Trp Leu Lys Pro
260 265 270
Gly Gly Gly Val Glu Asp Ser Ala Ala Gly Thr Pro Thr Ser Gly Val
275 280 285
Arg Ala Pro Gly Ile Pro Pro Ala Ser Leu Ser Leu Gln Ala Gly Gly
290 295 300
Leu Leu Gln His Pro His Gly Ala Ala Ala Gly Met Leu Gln Val Asp
305 310 315 320
Val Asp Asp Leu Tyr Arg Gly Gln Leu Ala Ala Ala Arg Gly Ala Ala
325 330 335
Leu Phe Ser Gly Gly Ile Asp Asp Val Gly Ser Val Tyr Ala Ala Gly
340 345 350
Ser Ala Gly Pro Ser Pro Thr Ala Leu Cys Ala Gly Arg Pro Ser Pro
355 360 365
Ser Pro Ser Pro Ser Ser Ser Thr Thr Ala Leu Ser Leu Leu Leu Arg
370 375 380
Ser Ser Val Phe Gln Glu Leu Val Ala Arg Asn Ala Gly Gly Gly Ala
385 390 395 400
Ala Gln Gln Gln Gln Leu Val Val Ala Asp Asp Asp Gly Ala Val Ser
405 410 415
Pro Ala Asp Val Val Asp Ala Lys Val Glu Gln Pro Glu Ala Glu Gly
420 425 430
Glu Leu Gly Arg His Gly Asp Gln Leu Tyr Gly Ala Ala Arg Ala Asp
435 440 445
Glu Asp Glu Asp Ala Phe Ala Cys Ser Met Tyr Glu Leu Asp Asp Ser
450 455 460
Phe Ala Arg Met Glu Gln Ser Leu Trp Gly Cys Leu Arg Ser Ser Asp
465 470 475 480
Ala Pro Asp Asn Met Asn Asn Leu
485
<210> 40
<211> 443
<212> БЕЛОК
<213> Sorghum bicolor
<400> 40
Met Glu Ser Ser Gly Met Met Met Val Lys Ser Glu Ile Glu Ser Cys
1 5 10 15
Gly Tyr Pro Gly Pro Ser Ser Ser Thr Ala Pro Ala Ala Gly Val Val
20 25 30
Ile Gly Gly Ser Ala Thr Thr Glu Arg Gly Glu Gly Gly His His His
35 40 45
His His His Gln Val Val Val Arg Arg Arg Arg Arg Glu Pro Pro Leu
50 55 60
Leu Ala Pro Ile Ala Gly Gly Gly Ile Gly Lys Pro Leu Pro Ser Ile
65 70 75 80
Thr Val Lys Arg Ser Ser Arg Phe Arg Gly Val Ser Arg His Arg Trp
85 90 95
Thr Gly Arg Phe Glu Ala His Leu Trp Asp Lys Asn Ser Trp Asn Pro
100 105 110
Thr Gln Arg Lys Lys Gly Lys Gln Val Tyr Leu Gly Ala Tyr Asp Glu
115 120 125
Glu Glu Ala Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp
130 135 140
Gly Pro Thr Thr Tyr Thr Asn Phe Pro Val Met Asp Tyr Glu Lys Glu
145 150 155 160
Leu Lys Ile Met Glu Asn Leu Thr Lys Glu Glu Tyr Leu Ala Ser Leu
165 170 175
Arg Arg Lys Ser Ser Gly Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly
180 185 190
Val Ala Arg His His Gln Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg
195 200 205
Val Phe Gly Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr Ser Thr Gln Glu
210 215 220
Glu Ala Ala Arg Ala Tyr Asp Ile Ala Ala Ile Glu Tyr Lys Gly Val
225 230 235 240
Asn Ala Val Thr Asn Phe Asp Leu Arg Ser Tyr Ile Thr Trp Leu Lys
245 250 255
Pro Ser Gly Ala Pro Ala Ala Phe Asn Pro Glu Ala Ala Leu Leu Met
260 265 270
Gln Ala Ala Pro Ala Glu Gln Leu Leu His Pro Ala Glu Thr Ala Gln
275 280 285
Met Leu Pro Arg Val Gly Asn Pro Phe Leu Leu Asp His Gly Ala Ala
290 295 300
Pro Pro Gly Ser Ser Gly Gly Gly Gly Gln Asp Ala Ser Met Ser Ser
305 310 315 320
Met Val Ser Pro Gly Ala Gly Gly Gly Met Arg Arg Arg Gly Ser Ser
325 330 335
Thr Ala Leu Ser Leu Leu Leu Lys Ser Ser Met Phe Arg Gln Leu Val
340 345 350
Glu Lys Asn Ser Asp Ala Glu Glu Gly Val Arg Asp Arg Glu Asp Ala
355 360 365
Ala Ala Ala Ala Ala Ala Ala His Pro Ala Gly Pro Gly Asp Ala Tyr
370 375 380
Glu Tyr His Asn Phe Phe Gln Gly Glu Ala Pro Pro Asp Met Cys Asp
385 390 395 400
Leu Phe Ser Ser Gly Gly Gly Gly Asp His Ala Arg Asn Ala Gly Phe
405 410 415
His Gly Glu Ile Ala Ala Cys Tyr Asp Asp Gly Glu Gly Leu Asp Gly
420 425 430
Trp Asn Gly Phe Gly Asn Met Ser Ser Leu Gln
435 440
<210> 41
<211> 407
<212> БЕЛОК
<213> Glycine max
<400> 41
Met Glu Leu Ala Pro Val Lys Ser Glu Leu Ser Pro Arg Ser His Arg
1 5 10 15
Leu Leu Met Ile Asp Gly Ser Glu Val Ile Gly Thr Lys Cys Val Lys
20 25 30
Arg Arg Arg Arg Asp Ser Ser Thr Ala Val Leu Gly Gly Asn Gly Gln
35 40 45
Gln Gly Glu Gln Leu Glu Glu Gln Lys Gln Leu Gly Gly Gln Ser Thr
50 55 60
Ala Thr Thr Val Lys Arg Ser Ser Arg Phe Arg Gly Val Ser Arg His
65 70 75 80
Arg Trp Thr Gly Arg Phe Glu Ala His Leu Trp Asp Lys Gly Thr Trp
85 90 95
Asn Pro Thr Gln Lys Lys Lys Gly Lys Gln Val Tyr Leu Gly Ala Tyr
100 105 110
Asn Asp Glu Glu Ala Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu Lys
115 120 125
Tyr Trp Gly Ile Ser Thr Phe Thr Asn Phe Pro Val Ser Asp Tyr Glu
130 135 140
Lys Glu Ile Glu Ile Met Lys Thr Val Thr Lys Glu Glu Tyr Leu Ala
145 150 155 160
Ser Leu Arg Arg Arg Ser Ser Gly Phe Ser Arg Gly Val Ser Lys Tyr
165 170 175
Arg Gly Val Ala Arg His His His Asn Gly Arg Trp Glu Ala Arg Ile
180 185 190
Gly Arg Val Phe Gly Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr Ser Thr
195 200 205
Gln Glu Glu Ala Ala Arg Ala Tyr Asp Ile Ala Ala Ile Glu Tyr Arg
210 215 220
Gly Ile Asn Ala Val Thr Asn Phe Asp Leu Ser Thr Tyr Ile Arg Trp
225 230 235 240
Leu Arg Pro Gly Thr His Pro Thr Ala Ser His Asp Gln Lys Pro Ser
245 250 255
Thr Asp Ala Gln Pro Phe Ala Thr Ser Asn Ser Met Gln Ala Arg Gly
260 265 270
Asn Ile Glu Val Ser Asn Ser Asn Lys Asn Ser Phe Pro Ser Gly Lys
275 280 285
Leu Asp Ser Thr Lys Lys Arg Asp Phe Ser Lys Tyr Met Asn Pro Leu
290 295 300
Ser Pro Cys Asn Lys Pro Ser Ser Pro Thr Ala Leu Gly Leu Leu Leu
305 310 315 320
Lys Ser Ser Val Phe Arg Glu Leu Met Gln Arg Asn Leu Asn Ser Ser
325 330 335
Ser Glu Glu Ala Glu Glu Val Glu Leu Lys Tyr Pro His Glu Gly Asn
340 345 350
Asp Gly Val Gly Gly Ile Tyr Asp Asn Glu Asn Thr Asn Asn Ser Tyr
355 360 365
Phe Cys Ser Ser Asn Ile Ser Arg Leu Pro Asn Leu Glu Ser Ser Glu
370 375 380
Glu Ser Pro Leu Pro Met Tyr His Gly Thr Val Gln Ser Leu Trp Asn
385 390 395 400
Ser Ala Phe Asn Met Ser Asn
405
<210> 42
<211> 406
<212> БЕЛОК
<213> Glycine max
<400> 42
Met Glu Leu Ala Pro Val Lys Ser Glu Leu Ser Pro Arg Ser His Arg
1 5 10 15
Leu Val Ile Ile Asp Gly Ser Asp Val Ile Ser Thr Lys Cys Ala Lys
20 25 30
Arg Arg Arg Arg Asp Ser Ser Met Ala Val Leu Gly Gly Asn Gly Gln
35 40 45
Gln Gly Glu Gln Leu Glu Glu Gln Lys Gln Leu Gly Gly Gln Ser Thr
50 55 60
Ala Thr Thr Val Lys Arg Ser Ser Arg Phe Arg Gly Val Ser Arg His
65 70 75 80
Arg Trp Thr Gly Arg Phe Glu Ala His Leu Trp Asp Lys Gly Thr Trp
85 90 95
Asn Pro Thr Gln Lys Lys Lys Gly Lys Gln Val Tyr Leu Gly Ala Tyr
100 105 110
Asn Asp Glu Glu Ala Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu Lys
115 120 125
Tyr Trp Gly Thr Ser Thr Phe Thr Asn Phe Pro Val Ser Asp Tyr Glu
130 135 140
Lys Glu Ile Glu Ile Met Lys Thr Val Thr Lys Glu Glu Tyr Leu Ala
145 150 155 160
Ser Leu Arg Arg Arg Ser Ser Gly Phe Ser Arg Gly Val Ser Lys Tyr
165 170 175
Arg Gly Val Ala Arg His His His Asn Gly Arg Trp Glu Ala Arg Ile
180 185 190
Gly Arg Val Phe Gly Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr Ser Thr
195 200 205
Gln Glu Glu Ala Ala Arg Ala Tyr Asp Ile Ala Ala Ile Glu Tyr Arg
210 215 220
Gly Ile Asn Ala Val Thr Asn Phe Asp Leu Ser Thr Tyr Ile Arg Trp
225 230 235 240
Leu Arg Pro Gly Thr His Pro Thr Ala Ser His Asp Gln Lys Pro Ser
245 250 255
Thr Asp Ala Gln Leu Phe Ala Thr Ser Asn Ser Met Gln Thr Arg Gly
260 265 270
Asn Ile Glu Val Ser Asn Ser Asn Met His Ser Phe Pro Ser Gly Glu
275 280 285
Leu Asp Ser Thr Lys Lys Arg Asp Phe Ser Lys Tyr Met Asn Pro Leu
290 295 300
Ser Pro Cys Asn Lys Pro Ser Ser Pro Thr Ala Leu Gly Leu Leu Leu
305 310 315 320
Lys Ser Ser Val Phe Arg Glu Leu Met Gln Arg Asn Leu Asn Ser Ser
325 330 335
Ser Glu Glu Ala Asp Val Glu Leu Lys Tyr Pro Gln Glu Gly Asn Asp
340 345 350
Gly Val Gly Gly Ile Tyr Asp Asn Asp Asn Thr Ser Asn Ser Tyr Phe
355 360 365
Cys Ser Ser Asn Ile Ser Arg Leu Pro Asn Leu Glu Ser Ser Glu Glu
370 375 380
Cys Pro Leu Pro Met Tyr His Gly Thr Met Gln Ser Leu Trp Asn Ser
385 390 395 400
Ala Phe Asn Met Ser Asn
405
<210> 43
<211> 418
<212> БЕЛОК
<213> Populus trichocarpa
<400> 43
Met Glu Met Thr Arg Asn Thr Gly Asp Gln Ile Ser Leu Gly Arg Arg
1 5 10 15
Arg Leu Cys Met Ile Glu Glu Glu Arg Arg Ala Gly Glu Ala Gly Lys
20 25 30
Cys Ile Lys Arg Arg Arg Arg Asp Pro Ser Thr Phe Ala Leu Ser Cys
35 40 45
Asn Ile Asn Asp Gln Gln Ser Asp Gln Gln Gln Gln Gln Gln Ser Leu
50 55 60
Gly Asp Arg Thr Ala Ala Val Ala Thr Thr Val Lys Arg Ser Ser Arg
65 70 75 80
Phe Arg Gly Val Ser Arg His Arg Trp Thr Gly Arg Phe Glu Ala His
85 90 95
Leu Trp Asp Lys Gly Thr Trp Asn Pro Thr Gln Arg Lys Lys Gly Lys
100 105 110
Gln Gly Ala Tyr Asp Glu Glu Glu Ser Ala Ala Arg Ala Tyr Asp Leu
115 120 125
Ala Ala Leu Lys Tyr Trp Gly Thr Ser Thr Phe Thr Asn Phe Pro Ala
130 135 140
Ser Asp Tyr Glu Lys Glu Ile Glu Ile Met Lys Thr Val Thr Lys Glu
145 150 155 160
Glu Tyr Leu Ala Ser Leu Arg Arg Arg Ser Ser Gly Phe Ser Arg Gly
165 170 175
Val Ser Lys Tyr Arg Gly Val Ala Arg His His His Asn Gly Arg Trp
180 185 190
Glu Ala Arg Ile Gly Arg Val Phe Gly Asn Lys Tyr Leu Tyr Leu Gly
195 200 205
Thr Tyr Ser Thr Gln Glu Glu Ala Ala His Ala Tyr Asp Ile Ala Ala
210 215 220
Ile Glu Tyr Arg Gly Ile Asn Ala Val Thr Asn Phe Asp Leu Ser Thr
225 230 235 240
Tyr Ile Arg Trp Leu Lys Pro Glu Ala Ser Leu Pro Ala Pro Gln Thr
245 250 255
Gln Glu Ser Lys Pro Ala Ser Asp Pro Leu Pro Met Ala Thr Phe Ser
260 265 270
Asn His Leu Pro Ser Glu Lys Pro Thr Gln Leu Ser Val Leu Gln Met
275 280 285
Asp Pro Ser Leu Met Asp Asn Leu Asn Thr Pro Lys Asn Glu Asp Ile
290 295 300
Phe His Arg Lys Thr Leu Pro Val Ser Pro Leu Thr Arg Ser Ser Ser
305 310 315 320
Ser Thr Ala Leu Ser Leu Leu Phe Lys Ser Ser Ile Phe Lys Glu Leu
325 330 335
Val Glu Lys Asn Leu Asn Thr Thr Ser Glu Glu Ile Glu Glu Asn Asp
340 345 350
Ser Lys Asn Pro His Asn Gly Asn Asn Asn Ala Gly Glu Ala Phe Tyr
355 360 365
Asp Gly Leu Ser Pro Ile Pro His Thr Gly Thr Ser Thr Glu Asp Pro
370 375 380
Phe Leu Cys Ser Glu Gln Gly Glu Thr Asn Thr Leu Pro Pro Tyr Ser
385 390 395 400
Gly Met Glu Gln Ser Leu Trp Asn Gly Ala Leu Ser Met Pro Ser Arg
405 410 415
Phe His
<210> 44
<211> 336
<212> БЕЛОК
<213> Vitis vinifera
<400> 44
Met Glu Met Thr Thr Val Lys Ser Glu Leu Gly Leu Glu Arg Gly Arg
1 5 10 15
Leu Cys Thr Ala Glu Thr Asp Ala Leu Glu Val Thr Lys Cys Val Lys
20 25 30
Arg Arg Arg Arg Asp Pro Ser Ala Val Thr Pro Gly Cys Ser Lys Gln
35 40 45
Gly Glu Gln Gln Lys Gln Val Leu Leu Gln Ala Gly Gln Ser Ile Thr
50 55 60
Ala Ile Ala Thr Thr Met Lys Arg Ser Ser Arg Phe Arg Gly Val Ser
65 70 75 80
Arg His Arg Trp Thr Gly Arg Phe Glu Ala His Leu Trp Asp Lys Gly
85 90 95
Ser Trp Asn Val Thr Gln Arg Lys Lys Gly Lys Gln Val Tyr Leu Gly
100 105 110
Ala Tyr Asp Glu Glu Glu Ser Ala Ala Arg Ala Tyr Asp Leu Ala Ala
115 120 125
Leu Lys Tyr Trp Gly Pro Ser Thr Phe Thr Asn Phe Pro Val Ser Asp
130 135 140
Tyr Glu Lys Glu Ile Glu Ile Met Gln Gly Leu Thr Lys Glu Glu Tyr
145 150 155 160
Leu Ala Ser Leu Arg Arg Arg Ser Ser Gly Phe Ser Arg Gly Val Ser
165 170 175
Lys Tyr Arg Gly Val Ala Arg His His His Asn Gly Arg Trp Glu Ala
180 185 190
Arg Ile Gly Arg Val Phe Gly Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr
195 200 205
Ser Thr Gln Glu Glu Ala Ala His Ala Tyr Asp Ile Ala Ala Ile Glu
210 215 220
Tyr Arg Gly Ile Asn Ala Val Thr Asn Phe Glu Leu Ser Thr Tyr Val
225 230 235 240
Arg Trp Leu Arg Pro Arg Ala Thr Ala Leu Thr Pro Gln Glu Pro Arg
245 250 255
Ser Asn Ser Ile Met Gln Ala Ser Ser Asn Cys Leu Pro Asn Glu Glu
260 265 270
Val Glu Leu Ser Phe Leu Ser Pro Asn Pro Phe Thr Val Asp Asp Leu
275 280 285
Ala Thr Pro Leu Lys Gln Glu Lys Phe Gln Arg Glu Val Ser Ile Ser
290 295 300
Pro Cys Thr Lys Ser Ser Ser Pro Thr Ala Leu Ser Leu Leu His Arg
305 310 315 320
Ser Ser Val Phe Arg Gln Leu Val Glu Lys Asn Ser Asn Ser Ile Glu
325 330 335
<210> 45
<211> 389
<212> БЕЛОК
<213> Glycine max
<400> 45
Met Ala Met Met Lys Glu Asn Ile Ile Glu Val Ser Leu Gly Arg Arg
1 5 10 15
Gln Met Ser Met Thr Glu Gly Glu Phe Gln Gly Thr Arg Ser Val Lys
20 25 30
Arg Arg Arg Arg Glu Val Ala Ala Ala Ala Gly Ser Gly Asp Asp Asn
35 40 45
His Gln Gln Gln Leu Pro Gln Gln Glu Val Gly Glu Asn Thr Thr Val
50 55 60
Asn Thr Thr Lys Arg Ser Ser Arg Phe Arg Gly Val Ser Arg His Arg
65 70 75 80
Trp Thr Gly Arg Tyr Glu Ala His Leu Trp Asp Lys Leu Ser Trp Asn
85 90 95
Ile Thr Gln Lys Lys Lys Gly Lys Gln Gly Ala Tyr Asp Glu Glu Glu
100 105 110
Ser Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Thr
115 120 125
Ser Thr Phe Thr Asn Phe Pro Ile Ser Asp Tyr Glu Lys Glu Ile Gln
130 135 140
Ile Met Gln Thr Met Thr Lys Glu Glu Tyr Leu Ala Thr Leu Arg Arg
145 150 155 160
Lys Ser Ser Gly Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly Val Ala
165 170 175
Arg His His His Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg Val Phe
180 185 190
Gly Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr Ser Thr Gln Glu Glu Ala
195 200 205
Ala Arg Ala Tyr Asp Ile Ala Ala Ile Glu Tyr Arg Gly Ile His Ala
210 215 220
Val Thr Asn Phe Asp Leu Ser Thr Tyr Ile Lys Trp Leu Lys Pro Ser
225 230 235 240
Gly Gly Gly Thr Leu Glu Ala Asn Leu Glu Ser His Ala Ala Leu Glu
245 250 255
His Gln Lys Val Ala Ser Pro Ser Asn Tyr Ala Leu Thr Glu Glu Ser
260 265 270
Lys Ser Leu Ala Leu His Asn Ser Phe Phe Ser Pro Tyr Ser Leu Asp
275 280 285
Ser Pro Val Lys His Glu Arg Phe Gly Asn Lys Thr Tyr Gln Phe Ser
290 295 300
Ser Asn Lys Ser Ser Ser Pro Thr Ala Leu Gly Leu Leu Leu Arg Ser
305 310 315 320
Ser Leu Phe Arg Glu Leu Val Glu Lys Asn Ser Asn Val Ser Gly Glu
325 330 335
Glu Asp Asp Gly Glu Ala Thr Lys Asp Gln Gln Thr Gln Ile Ala Thr
340 345 350
Asp Asp Asp Leu Gly Gly Ile Phe Phe Asp Ser Phe Ser Asp Ile Pro
355 360 365
Phe Val Cys Asp Pro Asn Arg Tyr Asp Leu Glu Leu Gln Glu Arg Asp
370 375 380
Leu His Ser Ile Phe
385
<210> 46
<211> 389
<212> БЕЛОК
<213> Glycine max
<400> 46
Met Val Met Met Lys Glu Asn Ile Ile Glu Glu Lys Leu Gly Arg Ser
1 5 10 15
Gln Met Ser Met Val Glu Gly Glu Phe Gln Gly Thr Trp Gly Val Lys
20 25 30
Arg Arg Arg Arg Glu Val Ala Ala Ala Ala Ser Ser Gly Asp Asp Asn
35 40 45
His His Gln Gln Leu Pro Gln Gln Glu Val Gly Glu Asn Ser Ser Ile
50 55 60
Ser Thr Thr Lys Arg Ser Ser Arg Phe Arg Gly Val Ser Arg His Arg
65 70 75 80
Trp Thr Gly Arg Tyr Glu Ala His Leu Trp Asp Lys Leu Ser Trp Asn
85 90 95
Ile Thr Gln Lys Lys Lys Gly Lys Gln Gly Ala Tyr Asp Glu Glu Glu
100 105 110
Ser Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Asn
115 120 125
Ser Thr Phe Thr Asn Phe Pro Ile Ser Asp Tyr Glu Lys Glu Ile Glu
130 135 140
Ile Met Gln Thr Met Thr Lys Glu Glu Tyr Leu Ala Thr Leu Arg Arg
145 150 155 160
Lys Ser Ser Gly Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly Val Ala
165 170 175
Arg His His His Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg Val Phe
180 185 190
Gly Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr Ser Thr Gln Glu Glu Ala
195 200 205
Ala Arg Ala Tyr Asp Ile Ala Ala Ile Glu Tyr Arg Gly Ile His Ala
210 215 220
Val Thr Asn Phe Asp Leu Ser Thr Tyr Ile Lys Trp Leu Lys Pro Ser
225 230 235 240
Gly Gly Gly Thr Pro Glu Glu Asn Leu Glu Ser His Ala Val Leu Glu
245 250 255
His Gln Lys Leu Ala Ser Pro Ser Asn Tyr Ala Leu Thr Glu Glu Ser
260 265 270
Lys Ser Leu Val Leu Pro Asn Ser Phe Ile Ser Pro Asp Ser Leu Asp
275 280 285
Ser Pro Val Lys His Glu Ser Phe Gly Asn Lys Thr Tyr Gln Phe Ser
290 295 300
Arg Asn Lys Ser Ser Ser Pro Thr Ala Leu Gly Leu Leu Leu Arg Ser
305 310 315 320
Ser Leu Phe Arg Glu Leu Val Glu Lys Asn Ser Asn Val Ser Gly Glu
325 330 335
Glu Ala Asp Gly Glu Val Thr Lys Asp Gln Gln Pro Gln Leu Ala Ser
340 345 350
Asp Asp Asp Leu Asp Gly Ile Phe Phe Asp Ser Phe Gly Asp Ile Pro
355 360 365
Phe Val Cys Asp Pro Thr Arg Tyr Asn Leu Glu Leu Gln Glu Arg Asp
370 375 380
Leu His Ser Ile Phe
385
<210> 47
<211> 392
<212> БЕЛОК
<213> Medicago truncatula
<400> 47
Met Ala Met Leu Ile Glu Asn Glu Val Met Cys Leu Gly Lys Ser Gln
1 5 10 15
Arg Ser Met Asp Gly Lys Glu Val Lys Gly Ala Arg Arg Val Lys Arg
20 25 30
Gln Arg Arg Asp Ala Ile Val Pro Lys Ile Gly Asp Asp Ala Asn Lys
35 40 45
Met Ala Gln Lys Gln Val Gly Glu Asn Ser Thr Thr Asn Thr Ser Lys
50 55 60
Arg Ser Ser Arg Phe Arg Gly Val Ser Arg His Arg Trp Thr Gly Arg
65 70 75 80
Phe Glu Ala His Leu Trp Asp Lys Leu Ser Trp Asn Thr Thr Gln Lys
85 90 95
Lys Lys Gly Lys Gln Gly Ala Tyr Asp Glu Glu Glu Ser Ala Ala Arg
100 105 110
Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Thr Ser Thr Phe Thr
115 120 125
Asn Phe Pro Ile Ser Asp Tyr Asp Lys Glu Ile Glu Ile Met Asn Thr
130 135 140
Met Thr Lys Glu Glu Tyr Leu Ala Thr Leu Arg Arg Lys Ser Ser Gly
145 150 155 160
Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly Val Ala Arg His His His
165 170 175
Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg Val Phe Gly Asn Lys Tyr
180 185 190
Leu Tyr Leu Gly Thr Tyr Ser Thr Gln Glu Glu Ala Ala Arg Ala Tyr
195 200 205
Asp Ile Ala Ala Ile Glu Tyr Arg Gly Ile His Ala Val Thr Asn Phe
210 215 220
Glu Leu Ser Ser Tyr Ile Lys Trp Leu Lys Pro Glu Thr Thr Thr Glu
225 230 235 240
Glu Asn His Glu Ser Gln Ile Leu Gln Lys Glu Ser Arg Thr Leu Ala
245 250 255
Pro Pro Asn Asn Ser Thr Leu Leu Gln Glu Ser Lys Leu Leu Ala Leu
260 265 270
Gln Lys Ser Phe Phe Ile Pro Asn Asp Leu Asn Ser Thr Glu Lys Gln
275 280 285
Glu Ser Ser Phe Glu Asn Lys Asn Tyr His Phe Leu Ser Asn Lys Ser
290 295 300
Thr Ser Pro Thr Ala Leu Ser Leu Leu Leu Arg Ser Ser Leu Phe Arg
305 310 315 320
Glu Leu Leu Glu Lys Asn Ser Asn Val Ser Glu Asp Glu Val Thr Lys
325 330 335
Glu Gln Gln Gln Gln Gln Ile Thr Ser Asp Asp Glu Leu Gly Gly Ile
340 345 350
Phe Tyr Asp Gly Ile Asp Asn Ile Ser Phe Asp Phe Asp Pro Asn Ser
355 360 365
Cys Asn Ile Glu Leu Gln Glu Arg Asp Leu His Ser Ile Ser Cys Leu
370 375 380
Tyr Gln Tyr Leu Asn Phe Gly Gln
385 390
<210> 48
<211> 386
<212> БЕЛОК
<213> Populus trichocarpa
<400> 48
Met Met Met Ile Lys Asn Glu Glu Asn Pro Gly Arg Arg Arg Gly Cys
1 5 10 15
Ile Ala Asp Ser Glu Ala Gln Val Ala Arg Cys Val Lys Arg Arg Arg
20 25 30
Arg Asp Pro Ala Ile Val Ala Leu Gly Ser Asp Asp Asn Gln Ser Gln
35 40 45
Gln Gln Met Pro Gln Lys Gln Thr Asp Gln Thr Ser Ala Ala Thr Thr
50 55 60
Val Lys Arg Ser Ser Arg Phe Arg Gly Val Ser Arg His Arg Trp Thr
65 70 75 80
Gly Arg Phe Glu Ala His Leu Trp Asp Lys Leu Ser Trp Asn Val Thr
85 90 95
Gln Lys Lys Lys Gly Lys Gln Gly Ala Tyr Asp Glu Glu Glu Ser Ala
100 105 110
Ala Arg Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Thr Ser Thr
115 120 125
Phe Thr Asn Phe Pro Ile Ser Asp Tyr Glu Lys Glu Ile Glu Ile Met
130 135 140
Gln Thr Val Thr Lys Glu Glu Tyr Leu Ala Ser Leu Arg Arg Lys Ser
145 150 155 160
Ser Gly Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly Val Ala Arg His
165 170 175
His His Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg Val Phe Gly Asn
180 185 190
Lys Tyr Leu Tyr Leu Gly Thr Tyr Ser Thr Gln Glu Glu Ala Ala Arg
195 200 205
Ala Tyr Asp Ile Ala Ala Ile Glu Tyr Arg Gly Ile Asn Ala Val Thr
210 215 220
Asn Phe Asp Leu Ser Thr Tyr Ile Arg Trp Ile Lys Pro Gly Val Ala
225 230 235 240
Ala Gln Ala Ala Ala Asn Glu Leu Gln Thr Val Thr Asp Pro Gln Thr
245 250 255
Ala Ala Thr Leu Thr Asp Thr Tyr Thr Pro Arg Glu Glu Thr Lys Pro
260 265 270
Ser Leu Phe Leu Pro Asn Gln Phe Thr Ala Asp Tyr Leu Asn Ser Pro
275 280 285
Pro Lys Leu Asp Ala Phe Gln Asn Asn Ile Phe Val Asp Ser Ser Asn
290 295 300
Lys Thr Ser Ser Pro Thr Ala Leu Ser Leu Leu Leu Arg Ser Ser Val
305 310 315 320
Phe Arg Glu Leu Val Glu Lys Asn Ser Asn Val Cys Glu Glu Glu Thr
325 330 335
Asp Gly Asn Glu Ile Lys Asn Gln Pro Met Ala Gly Ser Asp Asp Glu
340 345 350
Tyr Gly Gly Ile Phe Tyr Asp Gly Ile Gly Asp Ile Pro Phe Val Tyr
355 360 365
Ser Ser Asn Lys Tyr Ser Leu Gly Leu Glu Glu Arg Glu Leu Gln Phe
370 375 380
Val Leu
385
<210> 49
<211> 372
<212> БЕЛОК
<213> Ricinus communis
<400> 49
Met Glu Met Met Met Val Lys Asn Glu Glu Ile Ser Gly Arg Arg Arg
1 5 10 15
Ala Ser Val Thr Glu Ser Glu Ala Tyr Val Ala Arg Cys Val Lys Arg
20 25 30
Arg Arg Arg Asp Ala Ala Val Val Thr Val Gly Gly Asp Asp Ser Gln
35 40 45
Ser His Gln Gln Gln Gln Gln Gln Gln Pro Glu Gln Gln Ala His Gln
50 55 60
Ile Ser Ala Ala Thr Thr Val Lys Arg Ser Ser Arg Tyr Arg Gly Val
65 70 75 80
Ser Arg His Arg Trp Thr Gly Arg Phe Glu Ala His Leu Trp Asp Lys
85 90 95
Leu Ser Trp Asn Val Thr Gln Lys Lys Lys Gly Lys Gln Gly Ala Tyr
100 105 110
Asp Glu Glu Glu Ser Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu Lys
115 120 125
Tyr Trp Gly Thr Ser Thr Phe Thr Asn Phe Pro Ile Ser Asp Tyr Glu
130 135 140
Lys Glu Ile Glu Ile Met Gln Thr Val Thr Lys Glu Glu Tyr Leu Ala
145 150 155 160
Ser Leu Arg Arg Lys Ser Ser Gly Phe Ser Arg Gly Val Ser Lys Tyr
165 170 175
Arg Gly Val Ala Arg His His His Asn Gly Arg Trp Glu Ala Arg Ile
180 185 190
Gly Arg Val Phe Gly Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr Ser Thr
195 200 205
Gln Glu Glu Ala Ala Arg Ala Tyr Asp Ile Ala Ala Ile Glu Tyr Arg
210 215 220
Gly Ile Asn Ala Val Thr Asn Phe Asp Leu Ser Thr Tyr Ile Arg Trp
225 230 235 240
Leu Lys Pro Glu Val Ala Ala Gln Val Ala Ala Asn Glu Pro Gln Thr
245 250 255
Val Ala Glu Ser Arg Met Leu Pro Ser Ile Asn Asn Arg Ile Ala Arg
260 265 270
Glu Glu Ser Lys Pro Ser Phe Phe Ser Ala Thr Pro Phe Ser Leu Asp
275 280 285
Cys Trp Ser Tyr Pro Arg Lys Gln Glu Glu Phe Gln Asn Arg Thr Pro
290 295 300
Ile Thr Pro Cys Ser Lys Thr Ser Ser Pro Thr Ala Leu Ser Leu Leu
305 310 315 320
Leu Arg Ser Ser Ile Phe Arg Glu Leu Val Glu Lys Asn Ser Asn Val
325 330 335
Ser Glu Asp Glu Asn Glu Gly Glu Glu Thr Lys Asn Gln Ser Gln Ile
340 345 350
Gly Ser Asp Asp Glu Phe Gly Gly Leu Phe Tyr Glu Arg Ile Gly Asp
355 360 365
Ile Pro Phe Ile
370
<210> 50
<211> 404
<212> БЕЛОК
<213> Vitis vinifera
<400> 50
Met Glu Met Met Arg Val Lys Ser Glu Glu Asn Leu Gly Arg Arg Arg
1 5 10 15
Met Cys Val Ala Asp Ala Glu Ala Gln Gly Thr Arg Cys Val Lys Arg
20 25 30
Arg Arg Arg Asp Pro Ala Ile Val Thr Leu Gly Cys Asp Asp Gln Ser
35 40 45
Gln Gln Gln Gln Leu Pro Asn Gln Gln Pro Asp Gln Ala Ser Ala Ala
50 55 60
Thr Thr Val Lys Arg Ser Ser Arg Phe Arg Gly Val Ser Arg His Arg
65 70 75 80
Trp Thr Gly Arg Phe Glu Ala His Leu Trp Asp Lys Phe Ser Trp Asn
85 90 95
Val Thr Gln Lys Lys Lys Gly Lys Gln Gly Ala Tyr Asp Glu Glu Glu
100 105 110
Ser Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Ala
115 120 125
Ser Thr Phe Thr Asn Phe Pro Val Ser Asp Tyr Glu Lys Glu Ile Glu
130 135 140
Ile Met Gln Ser Val Thr Lys Glu Glu Tyr Leu Ala Cys Leu Arg Arg
145 150 155 160
Lys Ser Ser Gly Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly Val Ala
165 170 175
Arg His His His Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg Val Phe
180 185 190
Gly Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr Ser Thr Gln Glu Glu Ala
195 200 205
Ala Arg Ala Tyr Asp Ile Ala Ala Ile Glu Tyr Arg Gly Ile Asn Ala
210 215 220
Val Thr Asn Phe Asp Leu Ser Thr Tyr Ile Arg Trp Leu Asn Pro Ala
225 230 235 240
Ala Asn Asn Pro Val Val Pro His Glu Ser Arg Ala Asn Thr Glu Pro
245 250 255
Gln Ala Leu Ala Ser Ser Asn Phe Val Leu Ser Glu Glu Ser Glu Pro
260 265 270
Leu Phe Phe His Ser Asn Ser Phe Thr Met Asp Asp Leu Asn Pro Pro
275 280 285
His Lys Gln Glu Val Phe Gln Thr Lys Ile Pro Ile Glu Pro Cys Ser
290 295 300
Lys Ser Ser Ser Pro Thr Ala Leu Gly Leu Leu Leu Arg Ser Ser Ile
305 310 315 320
Phe Arg Glu Leu Val Glu Lys Asn Ser Asn Ala Pro Glu Asp Glu Thr
325 330 335
Asp Ala Glu Asp Thr Lys Asn Gln Gln Gln Val Gly Ser Asp Asp Glu
340 345 350
Tyr Gly Ile Phe Tyr Asp Gly Ile Gly Asp Ile Pro Phe Val Cys Pro
355 360 365
Ser Asn Gly Asp Arg Asn Glu Leu Gln Glu Arg Leu Pro Leu Pro Phe
370 375 380
Thr Ile Ser Gln Gly Asn Pro Tyr Gly Thr Ala Val Leu Thr Ser Met
385 390 395 400
Gln Ser Ile Asn
<210> 51
<211> 378
<212> БЕЛОК
<213> Brachypodium distachyon
<400> 51
Met Ala Lys Gln Arg Thr Asp Ser Ala Gly Thr Asp Ala Ala Ala Val
1 5 10 15
Gln Leu Thr Lys Pro Lys Arg Thr Arg Lys Ser Val Pro Arg Arg Glu
20 25 30
Ser Pro Ser Arg Arg Thr Ser Ala Tyr Arg Gly Val Thr Arg His Arg
35 40 45
Trp Thr Gly Arg Phe Glu Ala His Leu Trp Asp Lys Asn Thr Trp Thr
50 55 60
Gln Ser Gln Arg Lys Lys Lys Gly Arg Gln Val Tyr Leu Gly Ala Tyr
65 70 75 80
Gly Gly Glu Glu Ala Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu Lys
85 90 95
Tyr Trp Gly Arg Asp Thr Val Leu Asn Phe Pro Leu Ser Asn Tyr Asp
100 105 110
Glu Glu Trp Lys Glu Met Glu Gly Gln Ser Arg Glu Glu Tyr Ile Gly
115 120 125
Ser Leu Arg Arg Lys Ser Thr Gly Phe Ser Arg Gly Val Ser Lys Tyr
130 135 140
Arg Gly Val Ala Arg His His His Asn Gly Lys Trp Glu Ala Arg Ile
145 150 155 160
Gly Arg Val Tyr Gly Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr Gly Thr
165 170 175
Gln Glu Glu Ala Ala Met Ala Tyr Asp Ile Ala Ala Ile Glu His Arg
180 185 190
Gly Leu Asn Ala Val Thr Asn Phe Asp Val Ser Arg Tyr Ile Asp Trp
195 200 205
His Arg Arg Leu Cys Arg Asp Leu Gly Asp Asn Ile Ile Thr Pro Leu
210 215 220
Thr Asn Pro Thr Val Asp Leu Glu Glu Ala Met Ala Gly Asp Asp Asp
225 230 235 240
Asp Gly Gln Phe Leu Leu Pro Ser Gln Ala Thr Thr Pro Pro Ser Thr
245 250 255
Ser Ser Ala Leu Gly Leu Leu Leu Leu Ser Pro Arg Leu Lys Glu Val
260 265 270
Ile Glu Gly Ser Gly Ala Ala Ser Ala Met Ala Ala Ser Thr Ser Glu
275 280 285
Ser Ser Ala Ala Gly Ser Pro Pro Pro Ser Trp Ser Ser Ser Ser Cys
290 295 300
Ser Pro Ser Pro Pro Ser Pro Ser His Ser Pro Pro Glu Thr Gln Gln
305 310 315 320
Lys Gln Gln Gln Gln Glu Tyr Gly Ala Ser Ala Ala Ala Ala Arg Cys
325 330 335
Ser Phe Pro Asp Asp Val Gln Thr Tyr Phe Gly Cys Glu Asp Gly Cys
340 345 350
Ala Glu Val Asp Thr Phe Leu Phe Gly Asp Leu Ser Ala Tyr Ala Ala
355 360 365
Pro Met Phe Gln Phe Glu Leu Leu Asp Val
370 375
<210> 52
<211> 416
<212> БЕЛОК
<213> Oryza sativa
<400> 52
Met Ala Lys Arg Arg Ser Asn Gly Glu Thr Ala Ala Ala Ser Ser Asp
1 5 10 15
Asp Ser Ser Ser Gly Val Cys Gly Gly Gly Gly Gly Gly Glu Val Glu
20 25 30
Pro Arg Arg Arg Gln Lys Arg Pro Arg Arg Ser Ala Pro Arg Asp Cys
35 40 45
Pro Ser Gln Arg Ser Ser Ala Phe Arg Gly Val Thr Arg His Arg Trp
50 55 60
Thr Gly Arg Phe Glu Ala His Leu Trp Asp Lys Asn Thr Trp Asn Glu
65 70 75 80
Ser Gln Ser Lys Lys Gly Arg Gln Gly Ala Tyr Asp Gly Glu Glu Ala
85 90 95
Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly His Asp
100 105 110
Thr Val Leu Asn Phe Pro Leu Ser Thr Tyr Asp Glu Glu Leu Lys Glu
115 120 125
Met Glu Gly Gln Ser Arg Glu Glu Tyr Ile Gly Ser Leu Arg Arg Lys
130 135 140
Ser Ser Gly Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly Val Ala Arg
145 150 155 160
His His His Asn Gly Lys Trp Glu Ala Arg Ile Gly Arg Val Phe Gly
165 170 175
Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr Ala Thr Gln Glu Glu Ala Ala
180 185 190
Val Ala Tyr Asp Ile Ala Ala Ile Glu His Arg Gly Leu Asn Ala Val
195 200 205
Thr Asn Phe Asp Ile Asn Leu Tyr Ile Arg Trp Tyr His Gly Ser Cys
210 215 220
Arg Ser Ser Ser Ala Ala Ala Ala Thr Thr Ile Glu Asp Asp Asp Phe
225 230 235 240
Ala Glu Ala Ile Ala Ala Ala Leu Gln Gly Val Asp Glu Gln Pro Ser
245 250 255
Ser Ser Pro Ala Thr Thr Arg Gln Leu Gln Thr Ala Asp Asp Asp Asp
260 265 270
Asp Asp Leu Val Ala Gln Leu Pro Pro Gln Leu Arg Pro Leu Ala Arg
275 280 285
Ala Ala Ser Thr Ser Pro Ile Gly Leu Leu Leu Arg Ser Pro Lys Phe
290 295 300
Lys Glu Ile Ile Glu Gln Ala Ala Ala Ala Ala Ala Ser Ser Ser Gly
305 310 315 320
Ser Ser Ser Ser Ser Ser Thr Asp Ser Pro Ser Ser Ser Ser Ser Ser
325 330 335
Ser Leu Ser Pro Ser Pro Leu Pro Ser Pro Pro Pro Gln Gln Gln Pro
340 345 350
Thr Val Pro Lys Asp Asp Gln Tyr Asn Val Asp Met Ser Ser Val Ala
355 360 365
Ala Ala Arg Cys Ser Phe Pro Asp Asp Val Gln Thr Tyr Phe Gly Leu
370 375 380
Asp Asp Asp Gly Phe Gly Tyr Pro Glu Val Asp Thr Phe Leu Phe Gly
385 390 395 400
Asp Leu Gly Ala Tyr Ala Ala Pro Met Phe Gln Phe Glu Leu Asp Val
405 410 415
<210> 53
<211> 440
<212> БЕЛОК
<213> Sorghum bicolor
<400> 53
Met Ala Arg Pro Arg Lys Asn Ala Gly Thr Asp Glu Asp Asn Pro Asn
1 5 10 15
Ala Ala Thr Gly Val Ser Val Thr Gly Lys Pro Pro Lys Leu Lys Arg
20 25 30
Val Arg Arg Lys Gly Glu Pro Arg Glu Ser Ser Thr Pro Ser Gln Arg
35 40 45
Ser Ser Ala Tyr Arg Gly Val Thr Arg His Arg Trp Thr Gly Arg Phe
50 55 60
Glu Ala His Leu Trp Asp Lys Asp Ala Arg Asn Gly Ser Arg Asn Lys
65 70 75 80
Lys Gly Lys Gln Gly Ala Tyr Asp Asp Glu Glu Ala Ala Ala Arg Ala
85 90 95
His Asp Leu Ala Ala Leu Lys Tyr Trp Gly Pro Ala Thr Val Leu Asn
100 105 110
Phe Pro Leu Cys Gly Tyr Asp Glu Glu Leu Arg Glu Met Glu Ala Gln
115 120 125
Pro Arg Glu Glu Tyr Ile Gly Ser Leu Arg Arg Arg Ser Ser Gly Phe
130 135 140
Ser Arg Gly Val Ser Lys Tyr Arg Gly Val Ala Arg His His His Asn
145 150 155 160
Gly Arg Trp Glu Ala Arg Ile Gly Arg Val Leu Gly Asn Lys Tyr Leu
165 170 175
Tyr Leu Gly Thr Phe Ala Thr Gln Glu Glu Ala Ala Val Ala Tyr Asp
180 185 190
Ile Ala Ala Ile Glu His Arg Gly Leu Asn Ala Val Thr Asn Phe Asp
195 200 205
Ile Ser His Tyr Val Asn His Trp His Arg His Cys His Gly Pro Ser
210 215 220
Asp Asp Ser Leu Gly Val Val Val Asp Asp Val Ala Ala Phe Gln Leu
225 230 235 240
Pro Asp Asp Leu Pro Glu Cys Pro Ala Ala Ala Ile Gly Val Glu Glu
245 250 255
Thr Thr Gly Gly Asp Ala Glu Phe His Asn Gly Glu Glu Gly Tyr Leu
260 265 270
Gln His His Thr Ser Gly Pro Phe Gly Ala Gln Gln Gln Leu Pro Asp
275 280 285
Glu Thr Gly Ala Leu Ala Ala His Gln Met Ala Pro Asn Ser Ser Ala
290 295 300
Leu Asp Met Val Leu Gln Ser Pro Lys Phe Lys Glu Leu Met Glu Gln
305 310 315 320
Val Ser Ala Ala Ala Ala Ala Val Ala Ser Glu Ser Ser Ile Gly Gly
325 330 335
Ser Met Ser Ser Ser Ser Pro Ser Pro Ser Leu Ser Ser Phe Ser Pro
340 345 350
Ser Pro Leu Gln Leu Pro Ser Pro Ser Ser Leu Ser Ser Phe Ser Pro
355 360 365
Ser Ser Pro Leu Gln Gln Pro Ser Pro Pro Leu Gln Gln Pro Glu Phe
370 375 380
Val Glu Gly Ala Pro Ala Ala Arg Cys Ser Phe Pro Asp Asp Val Gln
385 390 395 400
Thr Phe Phe Asp Phe Glu Asn Glu Ser Asp Met Ser Phe Met Tyr Ala
405 410 415
Glu Val Asp Thr Phe Leu Phe Gly Asp Leu Gly Ala Tyr Ala Ala Pro
420 425 430
Ile Phe His Phe Asp Leu Asp Val
435 440
<210> 54
<211> 408
<212> БЕЛОК
<213> Zea mays
<400> 54
Met Ala Arg Pro Arg Lys Asn Gly Gly Thr Asp Glu Asp Asp Ala Asn
1 5 10 15
Ala Ala Thr Gly Ala Thr Gly Lys Pro Lys Lys Leu Met Lys Arg Ala
20 25 30
Arg Arg Lys Ser Glu Ser Pro Ser Pro Arg Ser Ser Ala Tyr Arg Gly
35 40 45
Val Thr Arg His Arg Trp Thr Gly Arg Phe Glu Ala His Leu Trp Asp
50 55 60
Lys Asp Ala Arg Asn Gly Ser Arg Ser Lys Lys Gly Lys Gln Val Tyr
65 70 75 80
Leu Gly Ala Tyr Asp Asp Glu Asp Ala Ala Ala Arg Ala His Asp Leu
85 90 95
Ala Ala Leu Lys Tyr Trp Gly Pro Ala Gly Thr Val Leu Asn Phe Pro
100 105 110
Leu Ser Gly Tyr Asp Glu Glu Arg Arg Glu Met Glu Gly Gln Pro Arg
115 120 125
Glu Glu Tyr Val Ala Ser Leu Arg Arg Arg Ser Ser Gly Phe Ala Arg
130 135 140
Gly Val Ser Lys Tyr Arg Gly Val Ala Arg His His His Asn Gly Arg
145 150 155 160
Trp Glu Ala Arg Ile Gly Arg Val Leu Gly Asn Lys Tyr Leu Tyr Leu
165 170 175
Gly Thr Tyr Ala Thr Gln Glu Glu Ala Ala Val Ala Tyr Asp Met Ala
180 185 190
Ala Ile Glu His Arg Gly Phe Asn Ala Val Thr Asn Phe Asp Ile Ser
195 200 205
His Tyr Ile Asn His Trp His Arg His Cys His Gly Pro Cys Asp Gly
210 215 220
Ser Leu Gly Ala Met Asp Val Ala Pro Asn Val Ser Leu Glu Leu Asp
225 230 235 240
Leu Leu Glu Cys Pro Ala Thr Val Gly Leu Gly Leu Glu Glu Thr Thr
245 250 255
Gly Asp Asp Glu Phe His Asn Arg Glu Asp Tyr Leu Gly His Leu Phe
260 265 270
Gly Val Gln Gln Leu Pro Asp Glu Met Gly Pro Pro Ala His Gln Met
275 280 285
Ala Pro Ala Ser Ser Ala Leu Asp Leu Val Leu Gln Ser Pro Arg Phe
290 295 300
Lys Glu Leu Met Gln Gln Val Ser Ala Ala Gly Ala Ser Glu Thr Asn
305 310 315 320
Gly Gly Ser Met Arg Ser Ser Pro Ser Thr Ser Leu Cys Ser Phe Ser
325 330 335
Pro Ser Pro Leu Glu Leu Pro Ser Pro Pro Leu Gln Gln Pro Thr Glu
340 345 350
Phe Ile Asp Gly Ala Pro Pro Arg Cys Ser Phe Pro Asp Asp Val Gln
355 360 365
Ser Phe Phe Asp Phe Lys Asn Asp Asn Asp Met Ser Phe Val Tyr Ala
370 375 380
Glu Val Asp Thr Phe Leu Phe Gly Asp Leu Gly Ala Tyr Ala Pro Pro
385 390 395 400
Met Phe Asp Phe Asp Leu Tyr Glu
405
<210> 55
<211> 304
<212> БЕЛОК
<213> Arabidopsis lyrata
<400> 55
Met Ala Lys Val Ser Arg Arg Ser Lys Lys Thr Ile Val Glu Asp Glu
1 5 10 15
Ile Ser Asp Lys Thr Ala Ser Ala Ser Glu Ala Ala Ser Ile Val Phe
20 25 30
Lys Ser Lys Arg Lys Arg Lys Ser Pro Pro Arg Asn Ala Pro Pro Gln
35 40 45
Arg Ser Ser Pro Tyr Arg Gly Val Thr Arg His Arg Trp Thr Gly Arg
50 55 60
Tyr Glu Ala His Leu Trp Asp Lys Asn Ser Trp Asn Glu Thr Gln Thr
65 70 75 80
Lys Lys Gly Arg Gln Val Tyr Ile Gly Ala Tyr Asp Glu Glu Glu Ala
85 90 95
Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Arg Asp
100 105 110
Thr Leu Leu Asn Phe Pro Leu Leu Ile Tyr Asp Glu Asp Val Lys Glu
115 120 125
Met Glu Gly Gln Ser Lys Glu Glu Tyr Ile Gly Ser Leu Arg Arg Lys
130 135 140
Ser Ser Gly Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly Val Ala Arg
145 150 155 160
His His His Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg Val Phe Gly
165 170 175
Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr Ala Thr Gln Glu Glu Ala Ala
180 185 190
Ile Ala Tyr Asp Ile Ala Ala Ile Glu Tyr Arg Gly Leu Asn Ala Val
195 200 205
Thr Asn Phe Asp Val Ser Arg Tyr Leu Asn Pro Asp Ala Ala Asp Ser
210 215 220
Lys Pro Ile Arg Asn Asp Pro Glu Ser Ser Asp Asp Asn Lys Cys Pro
225 230 235 240
Lys Ser Glu Glu Ile Ile Glu Pro Ser Thr Ser Pro Glu Ala Ile Thr
245 250 255
Thr Arg Arg Ser Phe Pro Asp Asp Ile Gln Thr Tyr Phe Gly Cys Gln
260 265 270
Asp Ser Gly Lys Leu Ala Thr Glu Glu Asp Val Ile Phe Gly Gly Leu
275 280 285
Asn Ser Phe Ile Asn Pro Gly Phe Tyr Asn Glu Phe Asp Tyr Gly Pro
290 295 300
<210> 56
<211> 308
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 56
Met Ala Lys Val Ser Gly Arg Ser Lys Lys Thr Ile Val Asp Asp Glu
1 5 10 15
Ile Ser Asp Lys Thr Ala Ser Ala Ser Glu Ser Ala Ser Ile Ala Leu
20 25 30
Thr Ser Lys Arg Lys Arg Lys Ser Pro Pro Arg Asn Ala Pro Leu Gln
35 40 45
Arg Ser Ser Pro Tyr Arg Gly Val Thr Arg Trp Thr Gly Arg Tyr Glu
50 55 60
Ala His Leu Trp Asp Lys Asn Ser Trp Asn Asp Thr Gln Thr Lys Lys
65 70 75 80
Gly Arg Gln Gly Ala Tyr Asp Glu Glu Glu Ala Ala Ala Arg Ala Tyr
85 90 95
Asp Leu Ala Ala Leu Lys Tyr Trp Gly Arg Asp Thr Leu Leu Asn Phe
100 105 110
Pro Leu Pro Ser Tyr Asp Glu Asp Val Lys Glu Met Glu Gly Gln Ser
115 120 125
Lys Glu Glu Tyr Ile Gly Ser Leu Arg Arg Lys Ser Ser Gly Phe Ser
130 135 140
Arg Gly Val Ser Lys Tyr Arg Gly Val Ala Arg His His His Asn Gly
145 150 155 160
Arg Trp Glu Ala Arg Ile Gly Arg Val Phe Gly Asn Lys Tyr Leu Tyr
165 170 175
Leu Gly Thr Tyr Ala Thr Gln Glu Glu Ala Ala Ile Ala Tyr Asp Ile
180 185 190
Ala Ala Ile Glu Tyr Arg Gly Leu Asn Ala Val Thr Asn Phe Asp Val
195 200 205
Ser Arg Tyr Leu Asn Pro Asn Ala Ala Ala Asp Lys Ala Asp Ser Asp
210 215 220
Ser Lys Pro Ile Arg Ser Pro Ser Arg Glu Pro Glu Ser Ser Asp Asp
225 230 235 240
Asn Lys Ser Pro Lys Ser Glu Glu Val Ile Glu Pro Ser Thr Ser Pro
245 250 255
Glu Val Ile Pro Thr Arg Arg Ser Phe Pro Asp Asp Ile Gln Thr Tyr
260 265 270
Phe Gly Cys Gln Asp Ser Gly Lys Leu Ala Thr Glu Glu Asp Val Ile
275 280 285
Phe Asp Cys Phe Asn Ser Tyr Ile Asn Pro Gly Phe Tyr Asn Glu Phe
290 295 300
Asp Tyr Gly Pro
305
<210> 57
<211> 303
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 57
Met Ala Lys Val Ser Gly Arg Ser Lys Lys Thr Ile Val Asp Asp Glu
1 5 10 15
Ile Ser Asp Lys Thr Ala Ser Ala Ser Glu Ser Ala Ser Ile Ala Leu
20 25 30
Thr Ser Lys Arg Lys Arg Lys Ser Pro Pro Arg Asn Ala Pro Leu Gln
35 40 45
Arg Ser Ser Pro Tyr Arg Gly Val Thr Arg His Arg Trp Thr Gly Arg
50 55 60
Tyr Glu Ala His Leu Trp Asp Lys Asn Ser Trp Asn Asp Thr Gln Thr
65 70 75 80
Lys Lys Gly Arg Gln Val Tyr Leu Gly Ala Tyr Asp Glu Glu Glu Ala
85 90 95
Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Arg Asp
100 105 110
Thr Leu Leu Asn Phe Pro Leu Pro Ser Tyr Asp Glu Asp Val Lys Glu
115 120 125
Met Glu Gly Gln Ser Lys Glu Glu Tyr Ile Gly Ser Leu Arg Arg Lys
130 135 140
Ser Ser Gly Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly Val Ala Arg
145 150 155 160
His His His Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg Val Phe Ala
165 170 175
Thr Gln Glu Glu Ala Ala Ile Ala Tyr Asp Ile Ala Ala Ile Glu Tyr
180 185 190
Arg Gly Leu Asn Ala Val Thr Asn Phe Asp Val Ser Arg Tyr Leu Asn
195 200 205
Pro Asn Ala Ala Ala Asp Lys Ala Asp Ser Asp Ser Lys Pro Ile Arg
210 215 220
Ser Pro Ser Arg Glu Pro Glu Ser Ser Asp Asp Asn Lys Ser Pro Lys
225 230 235 240
Ser Glu Glu Val Ile Glu Pro Ser Thr Ser Pro Glu Val Ile Pro Thr
245 250 255
Arg Arg Ser Phe Pro Asp Asp Ile Gln Thr Tyr Phe Gly Cys Gln Asp
260 265 270
Ser Gly Lys Leu Ala Thr Glu Glu Asp Val Ile Phe Asp Cys Phe Asn
275 280 285
Ser Tyr Ile Asn Pro Gly Phe Tyr Asn Glu Phe Asp Tyr Gly Pro
290 295 300
<210> 58
<211> 332
<212> БЕЛОК
<213> Arabidopsis lyrata
<400> 58
Met Glu Glu Ile Thr Arg Lys Ser Lys Lys Thr Ser Val Glu Asn Glu
1 5 10 15
Thr Gly Asp Asp Gln Ser Ala Thr Ser Val Val Val Lys Ala Lys Arg
20 25 30
Lys Arg Arg Ser Gln Pro Arg Asp Ala Pro Pro Gln Arg Ser Ser Val
35 40 45
His Arg Gly Val Thr Arg His Arg Trp Thr Gly Arg Tyr Glu Ala His
50 55 60
Leu Trp Asp Lys Asn Ser Trp Asn Glu Thr Gln Ser Lys Lys Gly Arg
65 70 75 80
Gln Gly Ala Tyr Asp Glu Glu Asp Ala Ala Ala Arg Ala Tyr Asp Leu
85 90 95
Ala Ala Leu Lys Tyr Trp Gly Arg Asp Thr Ile Leu Asn Phe Pro Leu
100 105 110
Cys Asn Tyr Glu Glu Asp Ile Lys Glu Met Glu Ser Gln Ser Lys Glu
115 120 125
Glu Tyr Ile Gly Ser Leu Arg Arg Lys Ser Ser Gly Phe Ser Arg Gly
130 135 140
Val Ser Lys Tyr Arg Gly Val Ala Lys His His His Asn Gly Arg Trp
145 150 155 160
Glu Ala Arg Ile Gly Arg Val Phe Gly Asn Lys Tyr Leu Tyr Leu Gly
165 170 175
Thr Tyr Ala Thr Gln Glu Glu Ala Ala Ile Ala Tyr Asp Ile Ala Ala
180 185 190
Ile Glu Tyr Arg Gly Leu Asn Ala Val Thr Asn Phe Asp Ile Ser Arg
195 200 205
Tyr Met Lys Leu Pro Val Pro Glu Asn Pro Ile Asp Ala Ala Asn Asn
210 215 220
Leu Leu Glu Ser Pro His Ser Asp Ser Ser Pro Phe Ile Asn Pro Thr
225 230 235 240
His Glu Ser Asp Leu Ser Gln Ser Gln Ser Ser Ser Asp Asp Asn Asp
245 250 255
Asp Arg Lys Thr Lys Leu Leu Lys Ser Ser Pro Leu Asn Ala Glu Glu
260 265 270
Val Ile Gly Pro Ser Thr Pro Pro Glu Ile Ala Pro Pro Arg Arg Ser
275 280 285
Phe Pro Glu Asp Ile Gln Thr Tyr Phe Gly Cys Gln Asn Ser Gly Lys
290 295 300
Leu Thr Thr Glu Glu Asp Asp Val Ile Phe Gly Asp Leu Asp Ser Phe
305 310 315 320
Leu Thr Pro Asp Phe Tyr Ser Glu Leu Asn Asp Cys
325 330
<210> 59
<211> 328
<212> БЕЛОК
<213> Thellungiella halophila
<400> 59
Met Ala Lys Val Ser Gln Arg Ser Lys Lys Thr Ile Val Asn Asp Glu
1 5 10 15
Ile Ser Asp Lys Lys Ala Val Ala Val Ala Ser Val Ser Ser Ser Ala
20 25 30
Phe Leu Lys Ser Lys Arg Lys Arg Lys Leu Pro Pro Gln Asn Ala Pro
35 40 45
Pro Gln Arg Ser Ser Ser Tyr Arg Gly Val Thr Arg His Arg Trp Thr
50 55 60
Gly Arg Tyr Glu Ala His Leu Trp Asp Lys Asn Cys Trp Asn Glu Thr
65 70 75 80
Gln Thr Lys Lys Gly Arg Gln Val Tyr Leu Gly Ala Tyr Asp Glu Glu
85 90 95
Glu Ala Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly
100 105 110
Arg Asp Thr Leu Leu Asn Phe Pro Leu Pro Thr Tyr Glu Glu Asp Val
115 120 125
Lys Glu Met Glu Gly His Ser Arg Glu Glu Tyr Ile Gly Ser Leu Arg
130 135 140
Arg Lys Ser Ser Gly Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly Val
145 150 155 160
Ala Arg His His His Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg Val
165 170 175
Phe Gly Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr Ala Thr Gln Glu Glu
180 185 190
Ala Ala Arg Ala Tyr Asp Ile Ala Ala Ile Glu Tyr Arg Gly Leu Asn
195 200 205
Ala Val Thr Asn Phe Asp Val Ser Arg Tyr Leu Asn Leu Pro Glu Ser
210 215 220
Lys Asn Pro Ser Ala Ala Ala Asn His Leu Pro Asp Glu Ser Asp Tyr
225 230 235 240
Tyr Asp Ser Met Pro Val Arg Asn Pro Asn His Glu Pro Arg Ser Pro
245 250 255
Asp Gly Gln Thr Ser Ser Glu Asp Asn Asp Tyr Thr Lys Thr Glu Glu
260 265 270
Thr Leu Asp Pro Glu Ala Ile Pro Ser Arg Arg Ser Phe Pro Asp Asp
275 280 285
Ile Gln Thr Tyr Phe Gly Cys Gln Asp Ser Gly Lys Leu Ala Thr Glu
290 295 300
Glu Asp Val Ile Phe Gly Gly Phe Asn Ser Phe Ile Asn Pro Gly Phe
305 310 315 320
Tyr Asn Asp Phe Asp Tyr Ala Pro
325
<210> 60
<211> 345
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 60
Met Phe Ile Ala Val Glu Val Ser Pro Val Met Glu Asp Ile Thr Arg
1 5 10 15
Gln Ser Lys Lys Thr Ser Val Glu Asn Glu Thr Gly Asp Asp Gln Ser
20 25 30
Ala Thr Ser Val Val Leu Lys Ala Lys Arg Lys Arg Arg Ser Gln Pro
35 40 45
Arg Asp Ala Pro Pro Gln Arg Ser Ser Val His Arg Gly Val Thr Arg
50 55 60
His Arg Trp Thr Gly Arg Tyr Glu Ala His Leu Trp Asp Lys Asn Ser
65 70 75 80
Trp Asn Glu Thr Gln Thr Lys Lys Gly Arg Gln Val Tyr Leu Gly Ala
85 90 95
Tyr Asp Glu Glu Asp Ala Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu
100 105 110
Lys Tyr Trp Gly Arg Asp Thr Ile Leu Asn Phe Pro Leu Cys Asn Tyr
115 120 125
Glu Glu Asp Ile Lys Glu Met Glu Ser Gln Ser Lys Glu Glu Tyr Ile
130 135 140
Gly Ser Leu Arg Arg Lys Ser Ser Gly Phe Ser Arg Gly Val Ser Lys
145 150 155 160
Tyr Arg Gly Val Ala Lys His His His Asn Gly Arg Trp Glu Ala Arg
165 170 175
Ile Gly Arg Val Phe Gly Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr Ala
180 185 190
Thr Gln Glu Glu Ala Ala Ile Ala Tyr Asp Ile Ala Ala Ile Glu Tyr
195 200 205
Arg Gly Leu Asn Ala Val Thr Asn Phe Asp Ile Ser Arg Tyr Leu Lys
210 215 220
Leu Pro Val Pro Glu Asn Pro Ile Asp Thr Ala Asn Asn Leu Leu Glu
225 230 235 240
Ser Pro His Ser Asp Leu Ser Pro Phe Ile Lys Pro Asn His Glu Ser
245 250 255
Asp Leu Ser Gln Ser Gln Ser Ser Ser Glu Asp Asn Asp Asp Arg Lys
260 265 270
Thr Lys Leu Leu Lys Ser Ser Pro Leu Val Ala Glu Glu Val Ile Gly
275 280 285
Pro Ser Thr Pro Pro Glu Ile Ala Pro Pro Arg Arg Ser Phe Pro Glu
290 295 300
Asp Ile Gln Thr Tyr Phe Gly Cys Gln Asn Ser Gly Lys Leu Thr Ala
305 310 315 320
Glu Glu Asp Asp Val Ile Phe Gly Asp Leu Asp Ser Phe Leu Thr Pro
325 330 335
Asp Phe Tyr Ser Glu Leu Asn Asp Cys
340 345
<210> 61
<211> 299
<212> БЕЛОК
<213> Glycine max
<400> 61
Met Ala Lys Lys Ser Gln Lys Ser Leu Lys Asn Asn Asn Asn Asn Asn
1 5 10 15
Thr Thr Arg Lys Arg Thr Arg Lys Ser Val Pro Arg Asp Ser Pro Pro
20 25 30
Gln Arg Ser Ser Ile Tyr Arg Gly Val Thr Arg His Arg Trp Thr Gly
35 40 45
Arg Tyr Glu Ala His Leu Trp Asp Lys Asn Cys Trp Asn Glu Ser Gln
50 55 60
Ser Lys Lys Gly Arg Gln Val Tyr Leu Gly Ala Tyr Asp Asp Glu Glu
65 70 75 80
Ala Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Gln
85 90 95
Asp Thr Ile Leu Asn Phe Pro Leu Ser Asn Tyr Glu Glu Lys Leu Lys
100 105 110
Glu Met Glu Gly Gln Ser Lys Glu Glu Tyr Ile Gly Ser Leu Arg Arg
115 120 125
Lys Ser Ser Gly Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly Val Ala
130 135 140
Arg His His His Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg Val Phe
145 150 155 160
Gly Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr Ala Thr Gln Glu Glu Ala
165 170 175
Ala Ala Ala Tyr Asp Met Ala Ala Ile Glu Tyr Arg Gly Leu Asn Ala
180 185 190
Val Thr Asn Phe Asp Leu Ser Arg Tyr Ile Asn Trp Pro Arg Pro Lys
195 200 205
Thr Glu Glu Asn His Gln Asn Thr Pro Ser Asn Gln Asn Val Asn Ser
210 215 220
Asn Ala Glu Leu Glu Leu Gly Ser Ala Ser Asp Glu Ile Thr Glu Glu
225 230 235 240
Gly Val Ala Arg Ser Ser Glu Ser Glu Ser Asn Pro Ser Arg Arg Thr
245 250 255
Phe Pro Glu Asp Ile Gln Thr Ile Phe Glu Asn Asn Gln Asp Ser Gly
260 265 270
Ile Tyr Ile Glu Asn Asp Asp Ile Ile Phe Gly Asp Leu Gly Ser Phe
275 280 285
Gly Ala Pro Ile Phe His Phe Glu Leu Asp Val
290 295
<210> 62
<211> 393
<212> БЕЛОК
<213> Brachypodium distachyon
<400> 62
Met Ala Lys Pro Arg Lys Asn Ser Ala Ala Ala Asn Asn Asn Asn Asn
1 5 10 15
Asp Asn Ser Thr Asn Ala Asn Asn Ala Val Ala Glu Ala Ala Ala Ala
20 25 30
Asp Val Arg Ala Lys Pro Lys Lys Arg Thr Arg Lys Ser Val Pro Arg
35 40 45
Glu Ser Pro Ser Gln Arg Ser Ser Ile Tyr Arg Gly Val Thr Arg His
50 55 60
Arg Trp Thr Gly Arg Phe Glu Ala His Leu Trp Asp Lys Asn Ser Trp
65 70 75 80
Asn Glu Ser Gln Asn Lys Lys Gly Lys Gln Val Tyr Leu Gly Ala Tyr
85 90 95
Asp Glu Glu Glu Ala Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu Lys
100 105 110
Tyr Trp Gly Pro Asp Thr Ile Leu Asn Phe Pro Leu Ser Val Tyr Asp
115 120 125
Asp Glu Leu Lys Glu Met Glu Gly Gln Ser Arg Glu Glu Tyr Ile Gly
130 135 140
Ser Leu Arg Arg Lys Ser Ser Gly Phe Ser Arg Gly Val Ser Lys Tyr
145 150 155 160
Arg Gly Val Ala Arg His His His Asn Gly Arg Trp Glu Ala Arg Ile
165 170 175
Gly Arg Val Phe Gly Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr Ala Thr
180 185 190
Gln Glu Glu Ala Ala Met Ala Tyr Asp Met Ala Ala Ile Glu Tyr Arg
195 200 205
Gly Leu Asn Ala Val Thr Asn Phe Asp Leu Ser Arg Tyr Ile Lys Trp
210 215 220
Leu Arg Pro Gly Gly Gly Val Asp Ser Ala Ala Ala Ala Ala Ala Arg
225 230 235 240
Asn Pro His Pro Met Leu Ala Gly Leu Ala Thr Gln Glu Glu Leu Pro
245 250 255
Ala Ile Asp His Leu Leu Asp Gly Met Ala Phe Gln Gln His Gly Leu
260 265 270
His Ser Ser Ser Ala Ala Ala Ala Ala Ala Gln Glu Phe Pro Leu Pro
275 280 285
Pro Ala Leu Gly His Ala Pro Thr Thr Ser Ala Leu Ser Leu Leu Leu
290 295 300
Gln Ser Pro Lys Phe Lys Glu Met Ile Glu Arg Thr Ser Ala Ala Glu
305 310 315 320
Thr Thr Thr Thr Ala Thr Thr Thr Ser Ser Ser Ser Ser Pro Arg Pro
325 330 335
Ala Ala Ser Pro Gln Cys Ser Phe Pro Glu Asp Ile Gln Thr Phe Phe
340 345 350
Gly Cys Asp Asp Gly Val Gly Val Gly Val Gly Ala Val Gly Tyr Thr
355 360 365
Asp Val Asp Gly Leu Phe Phe Gly Asp Leu Ser Ala Tyr Ala Ser Ser
370 375 380
Thr Ala Phe His Phe Glu Leu Asp Leu
385 390
<210> 63
<211> 379
<212> БЕЛОК
<213> Oryza sativa
<400> 63
Met Ala Lys Pro Arg Lys Asn Ser Thr Thr Thr Asn Thr Ser Ser Ser
1 5 10 15
Gly Val Ala Ala Ala Ala Ala Ala Ala Ala Val Lys Pro Lys Arg Thr
20 25 30
Arg Lys Ser Val Pro Arg Glu Ser Pro Ser Gln Arg Ser Ser Val Tyr
35 40 45
Arg Gly Val Thr Arg His Arg Trp Thr Gly Arg Phe Glu Ala His Leu
50 55 60
Trp Asp Lys Asn Ser Trp Asn Glu Ser Gln Asn Lys Lys Gly Lys Gln
65 70 75 80
Val Tyr Leu Gly Ala Tyr Asp Asp Glu Glu Ala Ala Ala Arg Ala Tyr
85 90 95
Asp Leu Ala Ala Leu Lys Tyr Trp Gly Pro Asp Thr Ile Leu Asn Phe
100 105 110
Pro Leu Ser Ala Tyr Glu Gly Glu Leu Lys Glu Met Glu Gly Gln Ser
115 120 125
Arg Glu Glu Tyr Ile Gly Ser Leu Arg Arg Lys Ser Ser Gly Phe Ser
130 135 140
Arg Gly Val Ser Lys Tyr Arg Gly Val Ala Arg His His His Asn Gly
145 150 155 160
Arg Trp Glu Ala Arg Ile Gly Arg Val Phe Gly Asn Lys Tyr Leu Tyr
165 170 175
Leu Gly Thr Tyr Ala Thr Gln Glu Glu Ala Ala Met Ala Tyr Asp Met
180 185 190
Ala Ala Ile Glu Tyr Arg Gly Leu Asn Ala Val Thr Asn Phe Asp Leu
195 200 205
Ser Arg Tyr Ile Lys Trp Leu Arg Pro Gly Ala Asp Gly Ala Gly Ala
210 215 220
Ala Gln Asn Pro His Pro Met Leu Gly Ala Leu Ser Ala Gln Asp Leu
225 230 235 240
Pro Ala Ile Asp Leu Asp Ala Met Ala Ser Ser Phe Gln His Asp Gly
245 250 255
His Gly Ala Ala Ala Ala Ala Ala Gln Leu Ile Pro Ala Arg His Ser
260 265 270
Leu Gly His Thr Pro Thr Thr Ser Ala Leu Ser Leu Leu Leu Gln Ser
275 280 285
Pro Lys Phe Lys Glu Met Ile Glu Arg Thr Ser Ala Ala Glu Thr Thr
290 295 300
Thr Thr Ser Ser Thr Thr Thr Ser Ser Ser Ser Pro Ser Pro Pro Gln
305 310 315 320
Ala Thr Lys Asp Asp Gly Ala Ser Pro Gln Cys Ser Phe Pro Lys Asp
325 330 335
Ile Gln Thr Tyr Phe Gly Cys Ala Ala Glu Asp Gly Ala Ala Gly Ala
340 345 350
Gly Tyr Ala Asp Val Asp Gly Leu Phe Phe Gly Asp Leu Thr Ala Tyr
355 360 365
Ala Ser Pro Ala Phe His Phe Glu Leu Asp Leu
370 375
<210> 64
<211> 398
<212> БЕЛОК
<213> Sorghum bicolor
<400> 64
Met Ala Lys Pro Arg Lys Asn Ser Ala Ala Ala Asn Asn Asn Asn Ser
1 5 10 15
Ser Ser Asn Gly Ala Gly Asp Leu Thr Pro Arg Ala Lys Pro Lys Arg
20 25 30
Thr Arg Lys Ser Val Pro Arg Glu Ser Pro Thr Gln Arg Ser Ser Val
35 40 45
Tyr Arg Gly Val Thr Arg His Arg Trp Thr Gly Arg Phe Glu Ala His
50 55 60
Leu Trp Asp Lys Asn Ser Trp Asn Glu Ser Gln Asn Lys Lys Gly Lys
65 70 75 80
Gln Val Tyr Leu Gly Ala Tyr Asp Asp Glu Glu Ala Ala Ala Arg Ala
85 90 95
Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Pro Asp Thr Ile Leu Asn
100 105 110
Phe Pro Ala Ser Ala Tyr Glu Gly Glu Met Lys Gly Met Glu Gly Gln
115 120 125
Ser Arg Glu Glu Tyr Ile Gly Ser Leu Arg Arg Lys Ser Ser Gly Phe
130 135 140
Ser Arg Gly Val Ser Lys Tyr Arg Gly Val Ala Arg His His His Asn
145 150 155 160
Gly Arg Trp Glu Ala Arg Ile Gly Arg Val Phe Gly Asn Lys Tyr Leu
165 170 175
Tyr Leu Gly Thr Tyr Ala Thr Gln Glu Glu Ala Ala Met Ala Tyr Asp
180 185 190
Met Ala Ala Ile Glu Tyr Arg Gly Leu Asn Ala Val Thr Asn Phe Asp
195 200 205
Leu Ser Arg Tyr Ile Lys Trp Leu Arg Pro Gly Ala Gly Gly Met Ala
210 215 220
Ala Ala Ala Ala Ala Ala Gln Asn Pro His Pro Met Leu Gly Gly Leu
225 230 235 240
Ala Gln Gln Leu Leu Leu Pro Pro Pro Ala Asp Thr Thr Thr Thr Asp
245 250 255
Gly Ala Gly Ala Ala Ala Phe Gln His Asp His His Gly Ala Glu Ala
260 265 270
Phe Pro Leu Pro Pro Arg Thr Ser Leu Gly His Thr Pro Thr Thr Ser
275 280 285
Ala Leu Ser Leu Leu Leu Gln Ser Pro Lys Phe Lys Glu Met Ile Gln
290 295 300
Arg Thr Glu Ser Gly Thr Thr Thr Thr Thr Thr Thr Thr Ser Ser Leu
305 310 315 320
Ser Ser Ser Pro Pro Pro Thr Pro Ser Pro Ser Pro Pro Arg Arg Ser
325 330 335
Pro Ala Pro Thr Gln Pro Pro Val Gln Ala Ala Ala Arg Asp Ala Ser
340 345 350
Pro His Gln Arg Gly Phe Pro Glu Asp Val Gln Thr Phe Phe Gly Cys
355 360 365
Glu Asp Thr Ala Gly Ile Asp Val Glu Ala Leu Phe Phe Gly Asp Leu
370 375 380
Ala Ala Tyr Ala Thr Pro Ala Phe His Phe Glu Met Asp Leu
385 390 395
<210> 65
<211> 396
<212> БЕЛОК
<213> Zea mays
<400> 65
Met Ala Arg Pro Arg Lys Asn Ser Ala Ala Ala Ala Asn Asn Asn Asn
1 5 10 15
Ser Asn Thr Thr Asn Ala Gly Asn Ala Ala Val Asp Leu Ala Ala Arg
20 25 30
Val Lys Pro Lys Arg Thr Arg Lys Ser Val Pro Arg Glu Ser Pro Ser
35 40 45
Gln Arg Ser Ser Val Tyr Arg Gly Val Thr Arg His Arg Trp Thr Gly
50 55 60
Arg Phe Glu Ala His Leu Trp Asp Lys Asn Ser Trp Asn Glu Ser Gln
65 70 75 80
Asn Lys Lys Gly Lys Gln Val Tyr Leu Gly Ala Tyr Asp Asp Glu Asp
85 90 95
Ala Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Pro
100 105 110
Asp Thr Ile Leu Asn Phe Pro Ala Ser Ala Tyr Glu Ala Glu Leu Lys
115 120 125
Glu Met Glu Gly Gln Ser Arg Glu Glu Tyr Ile Gly Ser Leu Arg Arg
130 135 140
Lys Ser Ser Gly Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly Val Ala
145 150 155 160
Arg His His His Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg Val Phe
165 170 175
Gly Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr Gly Thr Gln Glu Glu Ala
180 185 190
Ala Met Ala Tyr Asp Met Ala Ala Ile Glu Tyr Arg Gly Leu Asn Ala
195 200 205
Val Thr Asn Phe Asp Leu Ser Arg Tyr Ile Lys Trp Leu Arg Pro Gly
210 215 220
Ala Gly Ala Ala Gln Asn Pro His Pro Met Leu Asp Gly Leu Ala Gln
225 230 235 240
Gln Leu Leu Leu Ser Pro Glu Gly Thr Ile Asp Gly Ala Ala Phe His
245 250 255
Gln Gln Gln His Asp His Arg Gln Gln Gly Ala Ala Glu Leu Pro Leu
260 265 270
Pro Pro Arg Ala Ser Leu Gly His Thr Pro Thr Thr Ser Ala Leu Gly
275 280 285
Leu Leu Leu Gln Ser Ser Lys Phe Lys Glu Met Ile Gln Arg Ala Ser
290 295 300
Ala Ala Glu Ser Gly Thr Thr Thr Val Thr Thr Thr Ser Ser Ser Ser
305 310 315 320
Ser Gln Pro Pro Thr Pro Thr Pro Thr Pro Ser Pro Ser Pro Pro Pro
325 330 335
Thr Pro Pro Val Gln Pro Ala Arg Asp Ala Ser Pro Gln Cys Ser Phe
340 345 350
Pro Glu Asp Ile Gln Thr Phe Phe Gly Cys Glu Asp Val Ala Gly Val
355 360 365
Gly Ala Gly Val Asp Val Asp Ala Leu Phe Phe Gly Asp Leu Ala Ala
370 375 380
Tyr Ala Ser Pro Ala Phe His Phe Glu Met Asp Leu
385 390 395
<210> 66
<211> 362
<212> БЕЛОК
<213> Glycine max
<400> 66
Met Ala Lys Lys Ser Gln Leu Arg Thr Gln Lys Asn Asn Ala Thr Asn
1 5 10 15
Asp Asp Ile Asn Leu Asn Ala Thr Asn Thr Val Ile Thr Lys Val Lys
20 25 30
Arg Thr Arg Arg Ser Val Pro Arg Asp Ser Pro Pro Gln Arg Ser Ser
35 40 45
Ile Tyr Arg Gly Val Thr Arg His Arg Trp Thr Gly Arg Tyr Glu Ala
50 55 60
His Leu Trp Asp Lys His Cys Trp Asn Glu Ser Gln Asn Lys Lys Gly
65 70 75 80
Arg Gln Gly Ala Tyr Asp Asn Glu Glu Ala Ala Ala His Ala Tyr Asp
85 90 95
Leu Ala Ala Leu Lys Tyr Trp Gly Gln Asp Thr Ile Leu Asn Phe Pro
100 105 110
Leu Ser Asn Tyr Leu Asn Glu Leu Lys Glu Met Glu Gly Gln Ser Arg
115 120 125
Glu Glu Tyr Ile Gly Ser Leu Arg Arg Lys Ser Ser Gly Phe Ser Arg
130 135 140
Gly Ile Ser Lys Tyr Arg Gly Val Ala Arg His His His Asn Gly Arg
145 150 155 160
Trp Glu Ala Arg Ile Gly Lys Val Phe Gly Asn Lys Tyr Leu Tyr Leu
165 170 175
Gly Thr Tyr Ala Thr Gln Glu Glu Ala Ala Thr Ala Tyr Asp Leu Ala
180 185 190
Ala Ile Glu Tyr Arg Gly Leu Asn Ala Val Thr Asn Phe Asp Leu Ser
195 200 205
Arg Tyr Ile Lys Trp Leu Lys Pro Asn Asn Thr Asn Ser Asn Asn Asp
210 215 220
Gln Ile Ser Ile Asn Leu Thr Asn Ile Asn Asn Asn Cys Thr Asn Asn
225 230 235 240
Phe Ile Pro Asn Pro Asp Gln Glu Gln Glu Val Ser Phe Phe His Asn
245 250 255
Gln Asp Ser Leu Asn Asn Thr Ile Val Glu Glu Ala Thr Leu Val Pro
260 265 270
His Gln Pro Arg Pro Ala Ser Ala Thr Leu Ala Leu Glu Leu Leu Leu
275 280 285
Gln Ser Ser Lys Phe Lys Glu Met Val Glu Met Thr Ser Val Ala Asn
290 295 300
Leu Ser Thr Gln Met Glu Ser Asp Gln Leu Pro Gln Cys Thr Phe Pro
305 310 315 320
Asp His Ile Gln Thr Tyr Phe Glu Tyr Glu Asp Ser Asn Lys Tyr Glu
325 330 335
Glu Gly Asp Asp Leu Leu Phe Lys Phe Ser Glu Phe Ser Ser Ile Val
340 345 350
Pro Phe Tyr His Cys Asp Glu Phe Glu Ser
355 360
<210> 67
<211> 370
<212> БЕЛОК
<213> Glycine max
<400> 67
Met Ala Lys Lys Ser Gln Leu Arg Thr Gln Lys Asn Asn Val Thr Thr
1 5 10 15
Asn Asp Asp Asn Asn Leu Asn Val Thr Asn Thr Val Thr Thr Lys Val
20 25 30
Lys Arg Thr Arg Arg Ser Val Pro Arg Asp Ser Pro Pro Gln Arg Ser
35 40 45
Ser Ile Tyr Arg Gly Val Thr Arg His Arg Trp Thr Gly Arg Tyr Glu
50 55 60
Ala His Leu Trp Asp Lys His Cys Trp Asn Glu Ser Gln Asn Lys Lys
65 70 75 80
Gly Arg Gln Val Tyr Leu Gly Ala Tyr Asp Asn Glu Glu Ala Ala Ala
85 90 95
His Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Gln Asp Thr Ile
100 105 110
Leu Asn Phe Pro Leu Ser Asn Tyr Leu Asn Glu Leu Lys Glu Met Glu
115 120 125
Gly Gln Ser Arg Glu Glu Tyr Ile Gly Ser Leu Arg Arg Lys Ser Ser
130 135 140
Gly Phe Ser Arg Gly Ile Ser Lys Tyr Arg Gly Val Ala Arg His His
145 150 155 160
His Asn Gly Arg Trp Glu Ala Arg Ile Gly Lys Val Phe Gly Asn Lys
165 170 175
Tyr Leu Tyr Leu Gly Thr Tyr Ala Thr Gln Glu Glu Ala Ala Thr Ala
180 185 190
Tyr Asp Leu Ala Ala Ile Glu Tyr Arg Gly Leu Asn Ala Val Thr Asn
195 200 205
Phe Asp Leu Ser Arg Tyr Ile Lys Trp Leu Lys Pro Asn Asn Asn Thr
210 215 220
Asn Asn Val Ile Asp Asp Gln Ile Ser Ile Asn Leu Thr Asn Ile Asn
225 230 235 240
Asn Asn Asn Asn Cys Thr Asn Ser Phe Thr Pro Ser Pro Asp Gln Glu
245 250 255
Gln Glu Ala Ser Phe Phe His Asn Lys Asp Ser Leu Asn Asn Thr Ile
260 265 270
Val Glu Glu Val Thr Leu Val Pro His Gln Pro Arg Pro Ala Ser Ala
275 280 285
Thr Ser Ala Leu Glu Leu Leu Leu Gln Ser Ser Lys Phe Lys Glu Met
290 295 300
Met Glu Met Thr Ser Val Ala Asn Leu Ser Ser Thr Gln Met Glu Ser
305 310 315 320
Glu Leu Pro Gln Cys Thr Phe Pro Asp His Ile Gln Thr Tyr Phe Glu
325 330 335
Tyr Glu Asp Ser Asn Arg Tyr Glu Glu Gly Asp Asp Leu Met Phe Lys
340 345 350
Phe Asn Glu Phe Ser Ser Ile Val Pro Phe Tyr Gln Cys Asp Glu Phe
355 360 365
Glu Ser
370
<210> 68
<211> 356
<212> БЕЛОК
<213> Medicago truncatula
<400> 68
Met Ala Lys Lys Ser Gln Lys Gln Ile Glu Lys Asp Asp Asn Ala Ser
1 5 10 15
Asn Asp Asn Asp Asn Leu Asn Pro Ser Asn Thr Val Thr Thr Lys Ala
20 25 30
Lys Arg Thr Arg Lys Ser Val Pro Arg Thr Ser Pro Pro Gln Arg Ser
35 40 45
Ser Ile Tyr Arg Gly Val Thr Arg His Arg Trp Thr Gly Arg Tyr Glu
50 55 60
Ala His Leu Trp Asp Lys Asn Cys Trp Asn Glu Ser Gln Asn Lys Lys
65 70 75 80
Gly Arg Gln Gly Ala Tyr Asp Asn Glu Glu Thr Ala Ala His Ala Tyr
85 90 95
Asp Leu Ala Ala Leu Lys Tyr Trp Gly Gln Asp Thr Ile Ile Asn Phe
100 105 110
Pro Leu Ser Asn Tyr Gln Lys Glu Leu Ile Glu Met Glu Ser Gln Ser
115 120 125
Arg Glu Glu Tyr Ile Gly Ser Leu Arg Arg Lys Ser Ser Gly Phe Ser
130 135 140
Arg Gly Val Ser Lys Tyr Arg Gly Val Ala Arg His His His Asn Gly
145 150 155 160
Arg Trp Glu Ala Arg Ile Gly Lys Val Phe Gly Asn Lys Tyr Leu Tyr
165 170 175
Leu Gly Thr Tyr Ala Thr Gln Glu Glu Ala Ala Thr Ala Tyr Asp Met
180 185 190
Ala Ala Ile Glu Tyr Arg Gly Leu Asn Ala Val Thr Asn Phe Asp Leu
195 200 205
Ser Arg Tyr Ile Lys Trp Leu Lys Pro Asn Asn Asn Asn Asn Asp Asp
210 215 220
Asn Asn Lys Ser Asn Ile Asn Leu Cys Asp Ile Asn Ser Asn Ser Ser
225 230 235 240
Ala Asn Asp Ser Asn Ser Asn Glu Glu Leu Glu Phe Ser Leu Val Asp
245 250 255
Asn Glu Ile Ser Leu Asn Asn Ser Ile Asp Glu Ala Thr Leu Val Gln
260 265 270
Pro Arg Pro Thr Ser Ala Thr Ser Ala Leu Glu Leu Leu Leu Gln Ser
275 280 285
Ser Lys Phe Lys Glu Met Val Glu Met Ala Ser Met Thr Ser Asn Val
290 295 300
Ser Thr Thr Leu Glu Ser Asp Gln Leu Ser Gln Cys Ala Phe Pro Asp
305 310 315 320
Asp Ile Gln Thr Tyr Phe Glu Tyr Glu Asn Phe Asn Asp Thr Met Leu
325 330 335
Glu Asp Leu Asn Ser Ile Met Pro Thr Phe His Tyr Asp Phe Glu Gly
340 345 350
Ala Glu Val Leu
355
<210> 69
<211> 347
<212> БЕЛОК
<213> Glycine max
<400> 69
Met Ala Lys Gln Gln Thr His Lys Ile Asn Ala Ser Thr Asn Asn Asn
1 5 10 15
Ile Ser Thr Thr Asn Thr Val Thr Ala Lys Val Lys Arg Thr Arg Arg
20 25 30
Ser Val Pro Arg Asp Ser Pro Pro Gln Arg Ser Ser Ile Tyr Arg Gly
35 40 45
Val Thr Arg His Arg Trp Thr Gly Arg Tyr Glu Ala His Leu Trp Asp
50 55 60
Lys Asn Cys Trp Asn Glu Ser Gln Asn Lys Lys Gly Arg Gln Gly Ala
65 70 75 80
Tyr Asp Asp Glu Glu Ala Ala Ala His Ala Tyr Asp Leu Ala Ala Leu
85 90 95
Lys Tyr Trp Gly Gln Asp Thr Ile Leu Asn Phe Pro Leu Ser Thr Tyr
100 105 110
Gln Asn Glu Leu Lys Glu Met Glu Gly Gln Ser Arg Glu Glu Tyr Ile
115 120 125
Gly Ser Leu Arg Arg Lys Ser Ser Gly Phe Ser Arg Gly Val Ser Lys
130 135 140
Tyr Arg Gly Val Ala Arg His His His Asn Gly Arg Trp Glu Ala Arg
145 150 155 160
Ile Gly Arg Val Phe Gly Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr Ala
165 170 175
Thr Gln Glu Glu Ala Ala Thr Ala Tyr Asp Met Ala Ala Ile Glu Tyr
180 185 190
Arg Gly Val Asn Ala Val Thr Asn Phe Asp Leu Ser Arg Tyr Ile Lys
195 200 205
Trp Leu Lys Pro Asn Asn Asn Asn Thr Thr Val Asn Ser Asn Leu Ile
210 215 220
Asp Ser Asn Pro Asn Cys Glu Thr Asn Phe Thr Ser Asn Ser Asn Gln
225 230 235 240
Gln Gln Gly Phe Asn Phe Phe Asn Arg Gln Glu Ser Phe Asn Asn Glu
245 250 255
Glu Ala Ala Met Thr Gln Pro Arg Pro Ala Val Ala Thr Ser Ala Leu
260 265 270
Gly Leu Leu Leu Gln Ser Ser Lys Phe Lys Glu Met Met Glu Met Thr
275 280 285
Ser Ala Thr Asp Leu Ser Thr Pro Pro Ser Glu Ser Glu Leu Pro Ser
290 295 300
Cys Thr Phe Pro Asp Asp Ile Gln Thr Tyr Phe Glu Cys Glu Asp Ser
305 310 315 320
His Arg Tyr Gly Glu Gly Asp Asp Ile Met Phe Ser Val Leu Asn Gly
325 330 335
Phe Val Pro Pro Met Phe His Cys Asp Asp Phe
340 345
<210> 70
<211> 351
<212> БЕЛОК
<213> Glycine max
<400> 70
Met Ala Lys Gln Gln Thr His Glu Ile Asn Ala Ser Thr Asn Asn Asn
1 5 10 15
Ile Asn Thr Thr Lys Thr Val Thr Thr Lys Val Lys Arg Thr Arg Arg
20 25 30
Ser Val Pro Arg Asn Ser Pro Pro Gln Arg Ser Ser Ile Tyr Arg Gly
35 40 45
Val Thr Arg His Arg Trp Thr Gly Arg Tyr Glu Ala His Leu Trp Asp
50 55 60
Lys Asn Cys Trp Asn Glu Ser Gln Asn Lys Lys Gly Arg Gln Gly Ala
65 70 75 80
Tyr Asp Asp Glu Glu Ala Ala Ala His Ala Tyr Asp Leu Ala Ala Leu
85 90 95
Lys Tyr Trp Gly Gln Asp Thr Ile Leu Asn Phe Pro Leu Ser Thr Tyr
100 105 110
Gln Asn Glu Leu Lys Glu Met Glu Gly Gln Ser Arg Glu Glu Tyr Ile
115 120 125
Gly Ser Leu Arg Arg Lys Ser Ser Gly Phe Ser Arg Gly Val Ser Lys
130 135 140
Tyr Arg Gly Val Ala Arg His His His Asn Gly Arg Trp Glu Ala Arg
145 150 155 160
Ile Gly Arg Val Phe Gly Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr Ala
165 170 175
Thr Gln Glu Glu Ala Ala Thr Ala Tyr Asp Met Ala Ala Ile Glu Tyr
180 185 190
Arg Gly Leu Asn Ala Val Thr Asn Phe Asp Leu Ser Arg Tyr Ile Lys
195 200 205
Trp Leu Lys Pro Asn Asn Asn Asn Asn Lys Val Asn Ser Asn Asn Leu
210 215 220
Ile Val Ser Ile Pro Asn Cys Ala Thr Asn Phe Thr Pro Asn Ser Asn
225 230 235 240
Gln Gln Gln Gly Phe Asn Phe Phe Asn Ser Gln Glu Ser Phe Asn Asn
245 250 255
Asn Glu Glu Ala Ala Met Thr Gln Pro Arg Pro Ala Ala Ala Thr Ser
260 265 270
Ala Leu Gly Leu Leu Leu Gln Ser Ser Lys Phe Lys Glu Met Met Glu
275 280 285
Met Thr Ser Ala Ile Asp Leu Ser Thr Pro Pro Ser Glu Ser Glu Leu
290 295 300
Pro Pro Cys Thr Phe Pro Asp Asp Ile Gln Thr Tyr Phe Glu Cys Glu
305 310 315 320
Asp Ser His Arg Tyr Gly Glu Gly Asp Asp Ile Met Phe Ser Glu Leu
325 330 335
Asn Gly Phe Val Pro Pro Met Phe His Cys Asp Asp Phe Glu Ala
340 345 350
<210> 71
<211> 353
<212> БЕЛОК
<213> Populus trichocarpa
<400> 71
Met Ala Lys Leu Ser Gln Lys Asn Thr Lys Asn Thr Ala Ser Asn Asn
1 5 10 15
Asn Asn Thr Thr Asn Gly Val Thr Lys Val Lys Arg Thr Arg Arg Ser
20 25 30
Val Pro Arg Asp Ser Pro Pro Gln Arg Ser Ser Ile Tyr Arg Gly Val
35 40 45
Thr Arg His Arg Trp Thr Gly Arg Tyr Glu Ala His Leu Trp Asp Lys
50 55 60
Asn Cys Trp Asn Glu Ser Gln Asn Lys Lys Gly Arg Gln Gly Ala Tyr
65 70 75 80
Asp Asp Glu Glu Ala Ala Ala His Ala Tyr Asp Leu Ala Ala Leu Lys
85 90 95
Tyr Trp Gly Pro Glu Thr Ile Leu Asn Phe Pro Leu Ser Thr Tyr Gln
100 105 110
Asn Glu Leu Lys Glu Met Glu Gly Gln Ser Arg Glu Glu Cys Ile Gly
115 120 125
Ser Leu Arg Arg Lys Ser Ser Gly Phe Ser Arg Gly Val Ser Lys Tyr
130 135 140
Arg Gly Val Ala Arg His His His Asn Gly Arg Trp Glu Ala Arg Ile
145 150 155 160
Gly Arg Val Phe Gly Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr Ala Thr
165 170 175
Gln Glu Glu Ala Ala Thr Ala Tyr Asp Met Ala Ala Ile Glu Tyr Arg
180 185 190
Gly Leu Asn Ala Val Thr Asn Phe Asp Leu Ser Arg Tyr Ile Lys Trp
195 200 205
Leu Lys Pro Asn Gln Asn Asn Thr Asp Asn Asn Asn Gly Leu Asp Leu
210 215 220
Pro Asn Pro Ile Ile Gly Thr Asp Asn Ser Thr His Pro Asn Pro Asn
225 230 235 240
Gln Glu Leu Gly Thr Thr Phe Leu Gln Ile Asn Gln Gln Thr Tyr Gln
245 250 255
Pro Ser Glu Thr Thr Leu Thr Gln Pro Arg Pro Ala Thr Asn Pro Ser
260 265 270
Ser Ala Leu Gly Leu Leu Leu Gln Ser Ser Lys Phe Lys Glu Met Met
275 280 285
Glu Met Thr Ala Val Thr Asp Cys Pro Pro Thr Pro Pro Ser Gly Leu
290 295 300
Asp Pro Thr Pro Cys Ser Phe Leu Glu Asp Val Gln Thr Tyr Phe Asp
305 310 315 320
Cys Leu Asp Ser Ser Asn Tyr Gly Asp Gln Gly Asp Asp Met Ile Phe
325 330 335
Gly Asp Leu Asn Ser Phe Val Pro Pro Met Phe Gln Cys Asp Phe Glu
340 345 350
Thr
<210> 72
<211> 323
<212> БЕЛОК
<213> Vitis vinifera
<400> 72
Met Ala Lys Leu Ser Gln Gln Asn His Lys Asn Ser Ala Asn Ser Asn
1 5 10 15
Ala Thr Asn Thr Thr Leu Ser Val Thr Lys Val Lys Arg Thr Arg Lys
20 25 30
Thr Val Pro Arg Asp Ser Pro Pro Gln Arg Ser Ser Ile Tyr Arg Gly
35 40 45
Val Thr Arg His Arg Trp Thr Gly Arg Tyr Glu Ala His Leu Trp Asp
50 55 60
Lys Asn Cys Trp Asn Glu Ser Gln Asn Lys Lys Gly Arg Gln Val Tyr
65 70 75 80
Leu Gly Ala Tyr His Asp Glu Glu Ala Ala Ala His Ala Tyr Asp Leu
85 90 95
Ala Ala Leu Lys Tyr Trp Gly Pro Glu Thr Ile Leu Asn Phe Pro Leu
100 105 110
Ser Thr Tyr Glu Lys Glu Leu Lys Glu Met Glu Gly Leu Ser Arg Glu
115 120 125
Glu Tyr Ile Gly Ser Leu Arg Arg Arg Ser Ser Gly Phe Ser Arg Gly
130 135 140
Val Ser Lys Tyr Arg Gly Val Ala Arg His His His Asn Gly Arg Trp
145 150 155 160
Glu Ala Arg Ile Gly Arg Val Phe Gly Asn Lys Tyr Leu Tyr Leu Gly
165 170 175
Thr Tyr Ala Thr Gln Glu Glu Ala Ala Thr Ala Tyr Asp Met Ala Ala
180 185 190
Ile Glu Tyr Arg Gly Leu Asn Ala Val Thr Asn Phe Asp Leu Ser Arg
195 200 205
Tyr Ile Lys Trp Leu Lys Pro Asn Gln Asn Asn Pro Cys Glu Gln Pro
210 215 220
Asn Asn Pro Asn Leu Asp Ser Asn Leu Thr Pro Asn Pro Asn His Asp
225 230 235 240
Phe Gly Ile Ser Phe Leu Asn His Pro Gln Thr Ser Gly Thr Ala Ala
245 250 255
Cys Lys Met Met Glu Met Thr Thr Ala Ala Asp His Leu Ser Thr Pro
260 265 270
Pro Glu Ser Glu Leu Pro Arg Cys Ser Phe Pro Asp Asp Ile Gln Thr
275 280 285
Tyr Phe Glu Cys Gln Asp Ser Gly Ser Tyr Glu Glu Gly Asp Asp Val
290 295 300
Ile Phe Ser Glu Leu Asn Ser Phe Ile Pro Pro Met Phe Gln Cys Asp
305 310 315 320
Phe Ser Ala
<210> 73
<211> 347
<212> БЕЛОК
<213> Vitis vinifera
<400> 73
Met Ala Lys Leu Ser Gln Gln Asn His Lys Asn Ser Ala Asn Ser Asn
1 5 10 15
Ala Thr Asn Thr Thr Leu Ser Val Thr Lys Val Lys Arg Thr Arg Lys
20 25 30
Thr Val Pro Arg Asp Ser Pro Pro Gln Arg Ser Ser Ile Tyr Arg Gly
35 40 45
Val Thr Arg His Arg Trp Thr Gly Arg Tyr Glu Ala His Leu Trp Asp
50 55 60
Lys Asn Cys Trp Asn Glu Ser Gln Asn Lys Lys Gly Arg Gln Gly Ala
65 70 75 80
Tyr His Asp Glu Glu Ala Ala Ala His Ala Tyr Asp Leu Ala Ala Leu
85 90 95
Lys Tyr Trp Gly Pro Glu Thr Ile Leu Asn Phe Pro Leu Ser Thr Tyr
100 105 110
Glu Lys Glu Leu Lys Glu Met Glu Gly Leu Ser Arg Glu Glu Tyr Ile
115 120 125
Gly Ser Leu Arg Arg Arg Ser Ser Gly Phe Ser Arg Gly Val Ser Lys
130 135 140
Tyr Arg Gly Val Ala Arg His His His Asn Gly Arg Trp Glu Ala Arg
145 150 155 160
Ile Gly Arg Val Phe Gly Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr Ala
165 170 175
Thr Gln Glu Glu Ala Ala Thr Ala Tyr Asp Met Ala Ala Ile Glu Tyr
180 185 190
Arg Gly Leu Asn Ala Val Thr Asn Phe Asp Leu Ser Arg Tyr Ile Lys
195 200 205
Trp Leu Lys Pro Asn Gln Asn Asn Pro Cys Glu Gln Pro Asn Asn Pro
210 215 220
Asn Leu Asp Ser Asn Leu Thr Pro Asn Pro Asn His Asp Phe Gly Ile
225 230 235 240
Ser Phe Leu Asn His Pro Gln Thr Ser Gly Thr Ala Ala Cys Ser Glu
245 250 255
Pro Pro Leu Thr Gln Thr Arg Pro Pro Ile Ala Ser Ser Ala Leu Gly
260 265 270
Leu Leu Leu Gln Ser Ser Lys Phe Lys Glu Met Met Glu Met Thr Thr
275 280 285
Ala Ala Asp His Leu Ser Thr Pro Pro Glu Ser Glu Leu Pro Arg Cys
290 295 300
Ser Phe Pro Asp Asp Ile Gln Thr Tyr Phe Glu Cys Gln Asp Ser Gly
305 310 315 320
Ser Tyr Glu Glu Gly Asp Asp Val Ile Phe Ser Glu Leu Asn Ser Phe
325 330 335
Ile Pro Pro Met Phe Gln Cys Asp Phe Ser Ala
340 345
<210> 74
<211> 275
<212> БЕЛОК
<213> Populus trichocarpa
<400> 74
Met Gly Lys Thr Ser Lys Gln Ser Leu Lys Asn Ser Ala Asn Thr Ser
1 5 10 15
Ile Asn Pro Ala Thr Lys Val Lys Arg Thr Arg Lys Ser Val Pro Arg
20 25 30
Asp Ser Pro Pro Gln Arg Ser Ser Ile Tyr Arg Gly Val Thr Arg His
35 40 45
Arg Trp Thr Gly Arg Tyr Glu Ala His Leu Trp Asp Lys Asn Cys Trp
50 55 60
Asn Glu Ser Gln Asn Lys Lys Gly Arg Gln Gly Ala Tyr Asp Asp Glu
65 70 75 80
Glu Ala Ala Gly His Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly
85 90 95
Gln Asp Thr Ile Leu Asn Phe Pro Leu Ser Thr Tyr Glu Glu Glu Phe
100 105 110
Lys Glu Met Glu Gly His Ser Lys Glu Glu Tyr Ile Gly Ser Leu Arg
115 120 125
Arg Lys Ser Ser Gly Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly Val
130 135 140
Ala Arg His His His Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg Val
145 150 155 160
Phe Gly Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr Ala Thr Gln Glu Glu
165 170 175
Ala Ala Thr Ala Tyr Asp Met Ala Ala Ile Glu Tyr Arg Gly Leu Asn
180 185 190
Ala Val Thr Asn Phe Asp Leu Ser Arg Tyr Ser Ser Lys Phe Lys Glu
195 200 205
Met Leu Glu Arg Thr Ser Ala Ser Asp Cys Pro Leu Thr Pro Pro Glu
210 215 220
Ser Asp Arg Asp Pro Pro Arg Arg Ser Phe Pro Asp Asp Ile Gln Thr
225 230 235 240
Tyr Phe Asp Cys Gln Asp Ser Ser Ser Tyr Thr Asp Gly Asp Asp Ile
245 250 255
Ile Phe Gly Asp Leu His Ser Phe Ala Ser Pro Ile Phe His Cys Glu
260 265 270
Leu Asp Gly
275
<210> 75
<211> 304
<212> БЕЛОК
<213> Vitis vinifera
<400> 75
Met Ala Lys Thr Ser Gln Lys Ser Gln Lys Thr Thr Gly Asn Ser Thr
1 5 10 15
Asn Asn Asn Gly Gly Ser Val Ala Lys Val Lys Arg Thr Arg Lys Ser
20 25 30
Val Pro Arg Asp Ser Pro Pro Gln Arg Ser Ser Ile Phe Arg Gly Val
35 40 45
Thr Arg His Arg Trp Thr Gly Arg Tyr Glu Ala His Leu Trp Asp Lys
50 55 60
Asn Cys Trp Asn Glu Ser Gln Asn Lys Lys Gly Arg Gln Val Tyr Leu
65 70 75 80
Gly Ala Tyr Asp Asp Glu Glu Ala Ala Ala His Ala Tyr Asp Leu Ala
85 90 95
Ala Leu Lys Tyr Trp Gly Gln Glu Thr Ile Leu Asn Phe Pro Leu Ser
100 105 110
Ala Tyr Gln Glu Glu Leu Lys Glu Met Glu Gly Gln Ser Lys Glu Glu
115 120 125
Tyr Ile Gly Ser Leu Arg Arg Lys Ser Ser Gly Phe Ser Arg Gly Val
130 135 140
Ser Lys Tyr Arg Gly Val Ala Arg His His His Asn Gly Arg Trp Glu
145 150 155 160
Ala Arg Ile Gly Arg Val Phe Gly Asn Lys Tyr Leu Tyr Leu Gly Thr
165 170 175
Tyr Ala Thr Gln Glu Glu Ala Ala Thr Ala Tyr Asp Met Ala Ala Ile
180 185 190
Glu Tyr Arg Gly Leu Asn Ala Val Thr Asn Phe Asp Leu Ser Arg Tyr
195 200 205
Ile Asn Ser Pro Ala Pro Asn Pro Asn Pro Ser Asp His Glu Leu Gly
210 215 220
Leu Ser Phe Leu Gln Gln Gln His Gly Ser Asp Ala Thr Glu Leu Pro
225 230 235 240
Leu Ser His Ala Arg Ser Asp Cys Pro Leu Thr Pro Pro Asp Gln Ile
245 250 255
Glu Met Pro Arg Ser Ser Phe Pro Asp Asp Ile Gln Thr Tyr Phe Asp
260 265 270
Cys Gln Glu Thr Asn Ser Tyr Gly Glu Ser Asp Asp Ile Ile Phe Gly
275 280 285
Asp Leu Lys Tyr Phe Ser Ser Pro Met Phe Gln Cys Glu Leu Asp Thr
290 295 300
<210> 76
<211> 393
<212> БЕЛОК
<213> Sorghum bicolor
<400> 76
Met Ala Ser Pro Asn Pro Glu Ala Ala Ala Gly Leu Gln Thr Val Ala
1 5 10 15
Val Ala Ala Gly Gly Gly Glu Gly Gly Ser Ser Ser Ser Leu Gly Ala
20 25 30
Val Ala Gly Ala Ala Ala Val Ser Ser Ser Gly Glu Leu Val Pro Arg
35 40 45
Arg Ser Leu Ala Val Arg Lys Glu Arg Val Cys Thr Ala Lys Glu Arg
50 55 60
Ile Ser Arg Met Pro Pro Cys Ala Ala Gly Lys Arg Ser Ser Ile Tyr
65 70 75 80
Arg Gly Val Thr Arg His Arg Trp Thr Gly Arg Tyr Glu Ala His Leu
85 90 95
Trp Asp Lys Ser Thr Trp Asn Gln Asn Gln Asn Lys Lys Gly Lys Gln
100 105 110
Gly Ala Tyr Asp Asp Glu Glu Ala Ala Ala Arg Ala Tyr Asp Leu Ala
115 120 125
Ala Leu Lys Tyr Trp Gly Ala Gly Thr Gln Ile Asn Phe Pro Val Ser
130 135 140
Asp Tyr Ala Arg Asp Leu Glu Glu Met Gln Met Ile Ser Lys Glu Asp
145 150 155 160
Tyr Leu Val Ser Leu Arg Arg Gln Leu His Asn Ser Arg Trp Asp Thr
165 170 175
Ser Leu Gly Leu Gly Asn Asp Tyr Met Ser Leu Ser Cys Gly Lys Asp
180 185 190
Ile Met Leu Asp Gly Lys Phe Ala Gly Ser Phe Gly Leu Glu Arg Lys
195 200 205
Ile Asp Leu Thr Asn Tyr Ile Arg Trp Trp Leu Pro Lys Lys Thr Arg
210 215 220
Gln Ser Asp Thr Ser Lys Thr Glu Glu Ile Ala Asp Glu Ile Arg Ala
225 230 235 240
Ile Glu Ser Ser Met Gln Gln Thr Glu Pro Tyr Lys Leu Pro Ser Leu
245 250 255
Gly Leu Gly Ser Pro Ser Lys Pro Ser Ser Val Gly Leu Ser Ala Cys
260 265 270
Ser Ile Leu Ser Gln Ser Asp Ala Phe Lys Ser Phe Leu Glu Lys Ser
275 280 285
Thr Lys Leu Ser Glu Glu Cys Thr Leu Ser Lys Glu Ile Val Glu Gly
290 295 300
Lys Thr Val Ala Ser Val Pro Ala Thr Gly Tyr Asp Thr Gly Ala Ile
305 310 315 320
Asn Ile Asn Met Asn Glu Leu Leu Val Gln Arg Ser Thr Tyr Ser Met
325 330 335
Ala Pro Val Met Pro Thr Pro Met Lys Thr Thr Trp Ser Pro Ala Asp
340 345 350
Pro Ser Val Asp Pro Leu Phe Trp Ser Asn Phe Val Leu Pro Ser Ser
355 360 365
Gln Pro Val Thr Met Ala Thr Ile Thr Thr Thr Thr Asn Glu Val Ser
370 375 380
Ser Ser Asp Pro Phe Gln Ser Gln Glu
385 390
<210> 77
<211> 428
<212> БЕЛОК
<213> Lupinus angustifolius
<400> 77
Met Ala Ser Ser Ser Ser Asp Pro Gly Lys Ser Glu Ile Gly Gly Gly
1 5 10 15
Ala Ala Glu Thr Ser Glu Ala Ala Ala Val Ala Val Ala Val Thr Asn
20 25 30
Asp Gln Ser Leu Leu Tyr Arg Gly Leu Lys Lys Ala Lys Lys Glu Arg
35 40 45
Gly Cys Thr Ala Lys Glu Arg Ile Ser Lys Met Pro Pro Cys Ala Ala
50 55 60
Gly Lys Arg Ser Ser Ile Tyr Arg Gly Val Thr Arg His Arg Trp Thr
65 70 75 80
Gly Arg Tyr Glu Ala His Leu Arg Asp Lys Ser Thr Trp Asn Gln Asn
85 90 95
Gln Asn Lys Lys Gly Lys Gln Val Tyr Leu Gly Ala Tyr Asp Asp Glu
100 105 110
Glu Ala Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly
115 120 125
Pro Gly Thr Leu Ile Asn Phe Pro Val Thr Asp Tyr Thr Arg Asp Leu
130 135 140
Glu Glu Met Gln Asn Val Ser Arg Glu Glu Tyr Leu Ala Ser Leu Arg
145 150 155 160
Arg Lys Ser Ser Gly Phe Ser Arg Gly Ile Ser Lys Tyr Arg Ala Leu
165 170 175
Ser Ser Arg Trp Glu Pro Ser Tyr Ser Arg Phe Ala Gly Ser Asp Tyr
180 185 190
Phe Asn Ser Met His Tyr Gly Ala Gly Asp Asp Ser Ala Ala Glu Ser
195 200 205
Glu Tyr Ala Ser Gly Phe Cys Ile Glu Arg Lys Ile Asp Leu Thr Gly
210 215 220
His Ile Lys Trp Trp Gly Ser Asn Lys Ser Arg Gln Pro Asp Ala Gly
225 230 235 240
Thr Arg Leu Ser Glu Glu Lys Arg His Gly Phe Ala Gly Asp Ile Cys
245 250 255
Ser Glu Pro Lys Thr Leu Glu Gln Lys Val Gln Pro Thr Glu Pro Tyr
260 265 270
Gln Met Pro Glu Leu Gly Arg Ser His Asn Glu Lys Lys His Arg Ser
275 280 285
Ser Ala Val Ser Ala Leu Ser Ile Leu Ser Gln Ser Ala Ala Tyr Lys
290 295 300
Ser Leu Gln Glu Lys Ala Ser Lys Lys Gln Glu Asn Ser Thr Asp Asn
305 310 315 320
Asp Glu Asn Glu Asn Lys Asn Thr Val Asn Lys Leu Asp His Gly Lys
325 330 335
Ala Val Glu Lys Ser Ser Asn His Asp Gly Gly Ser Asp Arg Val Asp
340 345 350
Ile Glu Ile Gly Thr Thr Gly Ala Leu Ser Leu Gln Arg Asn Ile Tyr
355 360 365
Pro Leu Thr Pro Phe Leu Ser Ala Pro Leu Leu Thr Ala Tyr Asn Thr
370 375 380
Val Asp Pro Ser Leu Val Asp Pro Val Leu Trp Thr Ser Leu Val Pro
385 390 395 400
Met Leu Ser Ala Gly Leu Ser Cys Pro Thr Gln Val Thr Lys Thr Glu
405 410 415
Thr Ser Ser Ser Tyr Thr Ile Phe Gln Pro Glu Gly
420 425
<210> 78
<211> 440
<212> БЕЛОК
<213> Ricinus communis
<400> 78
Met Ala Ser Ser Ser Ser Asp Pro Gly Leu Lys Pro Glu Leu Gly Gly
1 5 10 15
Gly Ser Gly Gly Glu Ser Ser Glu Ala Val Ile Ala Asn Asp Gln Leu
20 25 30
Leu Leu Tyr Arg Gln Leu Lys Lys Pro Lys Lys Glu Arg Gly Cys Thr
35 40 45
Ala Lys Glu Arg Ile Ser Lys Met Pro Pro Cys Thr Ala Gly Lys Arg
50 55 60
Ser Ser Ile Tyr Arg Gly Val Thr Arg His Arg Trp Thr Gly Arg Tyr
65 70 75 80
Glu Ala His Leu Trp Asp Lys Ser Thr Trp Asn Gln Asn Gln Asn Lys
85 90 95
Lys Gly Lys Gln Gly Ala Tyr Asp Asp Glu Glu Ala Ala Ala Arg Ala
100 105 110
Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Pro Gly Thr Leu Ile Asn
115 120 125
Phe Pro Val Thr Asp Tyr Ser Arg Asp Leu Glu Glu Met Gln Asn Val
130 135 140
Ser Arg Glu Glu Tyr Leu Ala Ser Leu Arg Arg Lys Ser Ser Gly Phe
145 150 155 160
Ser Arg Gly Ile Ser Lys Tyr Arg Gly Leu Ser Ser Gln Trp Asp Ser
165 170 175
Ser Phe Gly Arg Met Pro Gly Ser Glu Tyr Phe Ser Ser Ile Asn Tyr
180 185 190
Gly Ala Ala Asp Asp Pro Ala Ala Glu Ser Glu Tyr Val Gly Ser Leu
195 200 205
Cys Phe Glu Arg Lys Ile Asp Leu Thr Ser Tyr Ile Arg Trp Trp Gly
210 215 220
Phe Asn Lys Thr Arg Glu Ser Val Ser Lys Ser Ser Asp Glu Arg Lys
225 230 235 240
His Gly Tyr Gly Glu Asp Ile Ser Glu Leu Lys Ser Ser Glu Trp Ala
245 250 255
Val Gln Ser Thr Glu Pro Tyr Gln Met Pro Arg Leu Gly Met Pro Asp
260 265 270
Asn Gly Lys Lys His Lys Cys Ser Lys Ile Ser Ala Leu Ser Ile Leu
275 280 285
Ser His Ser Ala Ala Tyr Lys Asn Leu Gln Glu Lys Ala Ser Lys Lys
290 295 300
Gln Glu Asn Cys Thr Asp Asn Asp Glu Lys Glu Asn Lys Lys Thr Asn
305 310 315 320
Lys Met Asp Tyr Gly Lys Ala Val Glu Lys Ser Thr Ser His Asp Gly
325 330 335
Ser Asn Glu Arg Leu Gly Ala Ala Leu Gly Met Ser Gly Gly Leu Ser
340 345 350
Leu Gln Arg Asn Ala Tyr Gln Leu Ala Pro Phe Leu Ser Ala Pro Leu
355 360 365
Leu Thr Asn Tyr Asn Ala Ile Asp Pro Leu Val Asp Pro Ile Leu Trp
370 375 380
Thr Ser Leu Val Pro Val Leu Pro Ala Gly Phe Ser Arg Asn Ser Glu
385 390 395 400
Val Gly Met Gly Leu Gln Ile Val Ser Cys His Lys Asp Arg Asp Lys
405 410 415
Phe Asn Leu Tyr Leu Leu Ser Ala Gly Gly Val Ser Thr Phe Leu Leu
420 425 430
Leu Val Val His Trp Arg Phe Cys
435 440
<210> 79
<211> 428
<212> БЕЛОК
<213> Lupinus angustifolius
<400> 79
Met Ala Ser Ser Ser Ser Asp Pro Gly Lys Ser Glu Ile Gly Gly Gly
1 5 10 15
Ala Ala Glu Thr Ser Glu Ala Ala Ala Val Ala Val Ala Val Thr Asn
20 25 30
Asp Gln Ser Leu Leu Tyr Arg Gly Leu Lys Lys Ala Lys Lys Glu Arg
35 40 45
Gly Cys Thr Ala Lys Glu Arg Ile Ser Lys Met Pro Pro Cys Ala Ala
50 55 60
Gly Lys Arg Ser Ser Ile Tyr Arg Gly Val Thr Arg His Arg Trp Thr
65 70 75 80
Gly Arg Tyr Glu Ala His Leu Arg Asp Lys Ser Thr Trp Asn Gln Asn
85 90 95
Gln Asn Lys Lys Gly Lys Gln Val Tyr Leu Gly Ala Tyr Asp Asp Glu
100 105 110
Glu Ala Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly
115 120 125
Pro Gly Thr Leu Ile Asn Phe Pro Val Thr Asp Tyr Thr Arg Asp Leu
130 135 140
Glu Glu Met Gln Asn Val Ser Arg Glu Glu Tyr Leu Ala Ser Leu Arg
145 150 155 160
Arg Lys Ser Ser Gly Phe Ser Arg Gly Ile Ser Lys Tyr Arg Ala Leu
165 170 175
Ser Ser Arg Trp Glu Pro Ser Tyr Ser Arg Phe Ala Gly Ser Asp Tyr
180 185 190
Phe Asn Ser Met His Tyr Gly Ala Gly Asp Asp Ser Ala Ala Glu Ser
195 200 205
Glu Tyr Ala Ser Gly Phe Cys Ile Glu Arg Lys Ile Asp Leu Thr Gly
210 215 220
His Ile Lys Trp Trp Gly Ser Asn Lys Ser Arg Gln Pro Asp Ala Gly
225 230 235 240
Thr Arg Leu Ser Glu Glu Lys Arg His Gly Phe Ala Gly Asp Ile Cys
245 250 255
Ser Glu Pro Lys Thr Leu Glu Gln Lys Val Gln Pro Thr Glu Pro Tyr
260 265 270
Gln Met Pro Glu Leu Gly Arg Ser His Asn Glu Lys Lys His Arg Ser
275 280 285
Ser Ala Val Ser Ala Leu Ser Ile Leu Ser Gln Ser Ala Ala Tyr Lys
290 295 300
Ser Leu Gln Glu Lys Ala Ser Lys Lys Gln Glu Asn Ser Thr Asp Asn
305 310 315 320
Asp Glu Asn Glu Asn Lys Asn Thr Val Asn Lys Leu Asp His Gly Lys
325 330 335
Ala Val Glu Lys Ser Ser Asn His Asp Gly Gly Ser Asp Arg Val Asp
340 345 350
Ile Glu Ile Gly Thr Thr Gly Ala Leu Ser Leu Gln Arg Asn Ile Tyr
355 360 365
Pro Leu Thr Pro Phe Leu Ser Ala Pro Leu Leu Thr Ala Tyr Asn Thr
370 375 380
Val Asp Pro Ser Leu Val Asp Pro Val Leu Trp Thr Ser Leu Val Pro
385 390 395 400
Met Leu Ser Ala Gly Leu Ser Cys Pro Thr Gln Val Thr Lys Thr Glu
405 410 415
Thr Ser Ser Ser Tyr Thr Ile Phe Gln Pro Glu Gly
420 425
<210> 80
<211> 508
<212> БЕЛОК
<213> Aspergillus fumigatus
<400> 80
Met Lys Met Ser Ala Ser Lys Thr Val Thr Ser Ser Ala Ser Ala Val
1 5 10 15
Ser Thr Ser Ser Gly Arg Ser Thr Pro Ser Lys Leu Val Asn Gly Ala
20 25 30
Thr Arg Asn Gly Ser Ala Ala Ala Gly Asn Gly Ser Thr Gly Thr Ala
35 40 45
Lys Gly Lys Arg Arg Ser Lys Tyr Arg His Val Ala Ala Tyr His Ser
50 55 60
Glu Leu Arg His Ser Ser Leu Ser Arg Glu Thr Ser Val Val Pro Ser
65 70 75 80
Phe Leu Gly Phe Arg Asn Leu Met Val Ile Val Leu Val Ala Met Asn
85 90 95
Leu Arg Leu Ile Ile Glu Asn Phe Met Lys Tyr Gly Val Leu Ile Cys
100 105 110
Ile Lys Cys His Asp Tyr Arg Lys Gln Asp Val Val Leu Gly Ser Ile
115 120 125
Leu Phe Ala Leu Val Pro Cys His Leu Phe Leu Ala Tyr Ile Ile Glu
130 135 140
Leu Val Ala Ala Gln Gln Ser Lys Lys Thr Val Gly Arg Gln Lys Lys
145 150 155 160
Asp Leu Ser Thr Glu Glu Arg Glu Arg Glu Gln Gln Ala Phe Arg Ser
165 170 175
Thr Trp Arg Tyr Thr Ala Phe Phe His Thr Val Asn Ala Thr Leu Cys
180 185 190
Leu Ala Val Thr Ser Phe Val Val Tyr Phe Tyr Ile Asn His Pro Gly
195 200 205
Ile Gly Thr Ile Cys Glu Leu His Ala Ile Ile Val Trp Leu Lys Asn
210 215 220
Cys Ser Tyr Ala Phe Thr Asn Arg Asp Leu Arg Gln Ala Met Val Asp
225 230 235 240
Pro Ser Ala Glu Ser Ala Leu Pro Glu Ile Tyr Ser Thr Cys Pro Tyr
245 250 255
Pro Arg Asn Ile Thr Leu Gly Asn Leu Thr Tyr Phe Trp Leu Ala Pro
260 265 270
Thr Leu Val Tyr Gln Pro Val Tyr Pro Arg Ser Ser His Ile Arg Trp
275 280 285
Ser Phe Val Ala Lys Arg Leu Ala Glu Phe Phe Gly Leu Ala Val Phe
290 295 300
Ile Trp Leu Leu Ser Ala Gln Tyr Ala Ala Pro Val Leu Arg Asn Ser
305 310 315 320
Ile Asp Lys Ile Ala Val Met Asp Ile Ala Ser Ile Leu Glu Arg Val
325 330 335
Met Lys Leu Ser Thr Ile Ser Leu Val Ile Trp Leu Ala Gly Phe Phe
340 345 350
Ala Leu Phe Gln Ser Leu Leu Asn Ala Leu Ala Glu Val Met Arg Phe
355 360 365
Gly Asp Arg Glu Phe Tyr Thr Asp Trp Trp Asn Ser Pro Ser Leu Gly
370 375 380
Ala Tyr Trp Arg Ser Trp Asn Arg Pro Val Tyr Leu Phe Met Lys Arg
385 390 395 400
His Val Phe Ser Pro Leu Val Gly Arg Gly Trp Ser Pro Phe Ala Ala
405 410 415
Ser Phe Met Val Phe Ser Leu Ser Ala Val Leu His Glu Met Leu Val
420 425 430
Gly Ile Pro Thr His Asn Leu Ile Gly Val Ala Phe Ala Gly Met Met
435 440 445
Phe Gln Leu Pro Leu Ile Ala Val Thr Ala Pro Phe Glu Lys Val Asn
450 455 460
Asp Ala Leu Gly Lys Ile Val Gly Asn Ser Ile Phe Trp Val Ser Phe
465 470 475 480
Cys Leu Val Gly Gln Pro Leu Gly Ala Leu Leu Tyr Phe Phe Ala Trp
485 490 495
Gln Ala Lys Tyr Gly Ser Val Ser Lys Ile His Val
500 505
<210> 81
<211> 521
<212> БЕЛОК
<213> Ricinus communis
<400> 81
Met Thr Ile Leu Glu Thr Pro Glu Thr Leu Gly Val Ile Ser Ser Ser
1 5 10 15
Ala Thr Ser Asp Leu Asn Leu Ser Leu Arg Arg Arg Arg Thr Ser Asn
20 25 30
Asp Ser Asp Gly Ala Leu Ala Asp Leu Ala Ser Lys Phe Asp Asp Asp
35 40 45
Asp Asp Val Arg Ser Glu Asp Ser Ala Glu Asn Ile Ile Glu Asp Pro
50 55 60
Val Ala Ala Val Thr Glu Leu Ala Thr Ala Lys Ser Asn Gly Lys Asp
65 70 75 80
Cys Val Ala Asn Ser Asn Lys Asp Lys Ile Asp Ser His Gly Gly Ser
85 90 95
Ser Asp Phe Lys Leu Ala Tyr Arg Pro Ser Val Pro Ala His Arg Ser
100 105 110
Leu Lys Glu Ser Pro Leu Ser Ser Asp Leu Ile Phe Lys Gln Ser His
115 120 125
Ala Gly Leu Phe Asn Leu Cys Ile Val Val Leu Val Ala Val Asn Ser
130 135 140
Arg Leu Ile Ile Glu Asn Leu Met Lys Tyr Gly Trp Leu Ile Lys Thr
145 150 155 160
Gly Phe Trp Phe Ser Ser Arg Ser Leu Arg Asp Trp Pro Leu Phe Met
165 170 175
Cys Cys Leu Ser Leu Pro Val Phe Pro Leu Ala Ala Tyr Leu Val Glu
180 185 190
Lys Ala Ala Tyr Arg Lys Tyr Ile Ser Pro Pro Ile Val Ile Phe Leu
195 200 205
His Val Ile Ile Thr Ser Ala Ala Val Leu Tyr Pro Ala Ser Val Ile
210 215 220
Leu Ser Cys Glu Ser Ala Phe Leu Ser Gly Val Thr Leu Met Glu Leu
225 230 235 240
Ala Cys Met Val Trp Leu Lys Leu Val Ser Tyr Ala His Thr Asn Tyr
245 250 255
Asp Met Arg Ala Ile Ala Asp Thr Ile His Lys Glu Asp Ala Ser Asn
260 265 270
Ser Ser Ser Thr Glu Tyr Cys His Asp Val Ser Phe Lys Thr Leu Ala
275 280 285
Tyr Phe Met Val Ala Pro Thr Leu Cys Tyr Gln Pro Ser Tyr Pro Arg
290 295 300
Thr Ala Phe Ile Arg Lys Gly Trp Val Phe Arg Gln Phe Val Lys Leu
305 310 315 320
Ile Ile Phe Thr Gly Phe Met Gly Phe Ile Ile Glu Gln Tyr Ile Asn
325 330 335
Pro Ile Val Gln Asn Ser Gln His Pro Leu Lys Gly Asp Leu Leu Tyr
340 345 350
Ala Ile Glu Arg Val Leu Lys Leu Ser Val Pro Asn Leu Tyr Val Trp
355 360 365
Leu Cys Leu Phe Tyr Cys Phe Phe His Leu Trp Leu Asn Ile Val Ala
370 375 380
Glu Leu Leu Arg Phe Gly Asp Arg Glu Phe Tyr Lys Asp Trp Trp Asn
385 390 395 400
Ala Lys Thr Val Glu Glu Tyr Trp Arg Met Trp Asn Met Pro Val His
405 410 415
Lys Trp Met Val Arg His Ile Tyr Phe Pro Cys Leu Arg Arg Lys Ile
420 425 430
Pro Arg Gly Val Ala Ile Val Ile Ala Phe Phe Val Ser Ala Val Phe
435 440 445
His Glu Leu Cys Ile Ala Val Pro Cys His Met Phe Lys Leu Trp Ala
450 455 460
Phe Phe Gly Ile Met Phe Gln Ile Pro Leu Val Val Ile Thr Asn Tyr
465 470 475 480
Phe Gln Arg Lys Phe Arg Ser Ser Met Val Gly Asn Met Ile Phe Trp
485 490 495
Phe Phe Phe Cys Ile Leu Gly Gln Pro Met Cys Val Leu Leu Tyr Tyr
500 505 510
His Asp Leu Met Asn Arg Asp Gly Asn
515 520
<210> 82
<211> 526
<212> БЕЛОК
<213> Vernicia fordii
<400> 82
Met Thr Ile Pro Glu Thr Pro Asp Asn Ser Thr Asp Ala Thr Thr Ser
1 5 10 15
Gly Gly Ala Glu Ser Ser Ser Asp Leu Asn Leu Ser Leu Arg Arg Arg
20 25 30
Arg Thr Ala Ser Asn Ser Asp Gly Ala Val Ala Glu Leu Ala Ser Lys
35 40 45
Ile Asp Glu Leu Glu Ser Asp Ala Gly Gly Gly Gln Val Ile Lys Asp
50 55 60
Pro Gly Ala Glu Met Asp Ser Gly Thr Leu Lys Ser Asn Gly Lys Asp
65 70 75 80
Cys Gly Thr Val Lys Asp Arg Ile Glu Asn Arg Glu Asn Arg Gly Gly
85 90 95
Ser Asp Val Lys Phe Thr Tyr Arg Pro Ser Val Pro Ala His Arg Ala
100 105 110
Leu Lys Glu Ser Pro Leu Ser Ser Asp Asn Ile Phe Lys Gln Ser His
115 120 125
Ala Gly Leu Phe Asn Leu Cys Ile Val Val Leu Val Ala Val Asn Ser
130 135 140
Arg Leu Ile Ile Glu Asn Ile Met Lys Tyr Gly Trp Leu Ile Lys Thr
145 150 155 160
Gly Phe Trp Phe Ser Ser Arg Ser Leu Arg Asp Trp Pro Leu Leu Met
165 170 175
Cys Cys Leu Thr Leu Pro Ile Phe Ser Leu Ala Ala Tyr Leu Val Glu
180 185 190
Lys Leu Ala Cys Arg Lys Tyr Ile Ser Ala Pro Thr Val Val Phe Leu
195 200 205
His Ile Leu Phe Ser Ser Thr Ala Val Leu Tyr Pro Val Ser Val Ile
210 215 220
Leu Ser Cys Glu Ser Ala Val Leu Ser Gly Val Ala Leu Met Leu Phe
225 230 235 240
Ala Cys Ile Val Trp Leu Lys Leu Val Ser Tyr Ala His Thr Asn Phe
245 250 255
Asp Met Arg Ala Ile Ala Asn Ser Val Asp Lys Gly Asp Ala Leu Ser
260 265 270
Asn Ala Ser Ser Ala Glu Ser Ser His Asp Val Ser Phe Lys Ser Leu
275 280 285
Val Tyr Phe Met Val Ala Pro Thr Leu Cys Tyr Gln Pro Ser Tyr Pro
290 295 300
Arg Thr Ala Ser Ile Arg Lys Gly Trp Val Val Arg Gln Phe Val Lys
305 310 315 320
Leu Ile Ile Phe Thr Gly Phe Met Gly Phe Ile Ile Glu Gln Tyr Ile
325 330 335
Asn Pro Ile Val Gln Asn Ser Gln His Pro Leu Lys Gly Asp Leu Leu
340 345 350
Tyr Ala Ile Glu Arg Val Leu Lys Leu Ser Val Pro Asn Leu Tyr Val
355 360 365
Trp Leu Cys Met Phe Tyr Cys Phe Phe His Leu Trp Leu Asn Ile Leu
370 375 380
Ala Glu Leu Leu Arg Phe Gly Asp Arg Glu Phe Tyr Lys Asp Trp Trp
385 390 395 400
Asn Ala Arg Thr Val Glu Glu Tyr Trp Arg Met Trp Asn Met Pro Val
405 410 415
His Lys Trp Met Val Arg His Ile Tyr Phe Pro Cys Leu Arg His Lys
420 425 430
Ile Pro Arg Gly Val Ala Leu Leu Ile Thr Phe Phe Val Ser Ala Val
435 440 445
Phe His Glu Leu Cys Ile Ala Val Pro Cys His Ile Phe Lys Leu Trp
450 455 460
Ala Phe Ile Gly Ile Met Phe Gln Ile Pro Leu Val Gly Ile Thr Asn
465 470 475 480
Tyr Leu Gln Asn Lys Phe Arg Ser Ser Met Val Gly Asn Met Ile Phe
485 490 495
Trp Phe Ile Phe Cys Ile Leu Gly Gln Pro Met Cys Leu Leu Leu Tyr
500 505 510
Tyr His Asp Leu Met Asn Arg Lys Gly Thr Thr Glu Ser Arg
515 520 525
<210> 83
<211> 523
<212> БЕЛОК
<213> Vernonia galamensis
<400> 83
Met Ala Leu Leu Asp Thr Pro Gln Ile Gly Glu Ile Thr Thr Thr Ala
1 5 10 15
Thr Thr Thr Ile Arg Arg Arg Thr Thr Val Lys Pro Asp Ala Gly Ile
20 25 30
Gly Asp Gly Leu Phe Asp Ser Ser Ser Ser Ser Lys Thr Asn Ser Ser
35 40 45
Phe Glu Asp Gly Asp Ser Leu Asn Gly Asp Phe Asn Asp Lys Phe Lys
50 55 60
Glu Gln Ile Gly Ala Gly Asp Glu Ser Lys Asp Asp Ser Lys Gly Asn
65 70 75 80
Gly Gln Lys Ile Asp His Gly Gly Val Lys Lys Gly Arg Glu Thr Thr
85 90 95
Val Val His Tyr Ala Tyr Arg Pro Ser Ser Pro Ala His Arg Arg Ile
100 105 110
Lys Glu Ser Pro Leu Ser Ser Asp Ala Ile Phe Lys Gln Ser His Ala
115 120 125
Gly Leu Phe Asn Leu Cys Ile Val Val Leu Val Ala Val Asn Gly Arg
130 135 140
Leu Ile Ile Glu Asn Leu Met Lys Tyr Gly Leu Leu Ile Asn Ser Asn
145 150 155 160
Phe Trp Phe Ser Ser Arg Ser Leu Arg Asp Trp Pro Leu Leu Met Cys
165 170 175
Cys Leu Thr Pro Ser Asp Phe Pro Leu Ala Ala Tyr Ile Val Glu Lys
180 185 190
Leu Ala Trp Lys Lys Arg Ile Ser Asp Pro Val Val Ile Thr Leu His
195 200 205
Val Ile Ile Thr Thr Thr Ala Ile Leu Tyr Pro Val Phe Met Ile Leu
210 215 220
Arg Phe Asp Ser Val Val Leu Ser Gly Val Ser Leu Met Leu Cys Ala
225 230 235 240
Cys Ile Asn Trp Leu Lys Leu Val Ser Phe Val His Thr Asn Tyr Asp
245 250 255
Met Arg Ser Leu Leu Asn Ser Thr Asp Lys Gly Glu Val Glu Pro Met
260 265 270
Ser Ser Asn Met Asp Tyr Phe Tyr Asp Val Asn Phe Lys Ser Leu Val
275 280 285
Tyr Phe Met Val Ala Pro Thr Leu Cys Tyr Gln Ile Ser Tyr Pro Arg
290 295 300
Thr Ala Phe Ile Arg Lys Gly Trp Val Leu Arg Gln Leu Ile Lys Leu
305 310 315 320
Val Ile Phe Thr Gly Phe Met Gly Phe Ile Ile Glu Gln Tyr Ile Asn
325 330 335
Pro Ile Val Lys Asn Ser Arg His Pro Leu Lys Gly Asp Phe Leu Tyr
340 345 350
Ala Ile Glu Arg Val Leu Lys Leu Ser Val Pro Asn Leu Tyr Val Trp
355 360 365
Leu Cys Met Phe Tyr Cys Phe Phe His Leu Trp Leu Asn Ile Leu Ala
370 375 380
Glu Leu Leu Cys Phe Gly Asp Arg Glu Phe Tyr Lys Asp Trp Trp Asn
385 390 395 400
Ala Gln Thr Ile Glu Glu Tyr Trp Arg Leu Trp Asn Met Pro Val His
405 410 415
Lys Trp Ile Val Arg His Leu Tyr Phe Pro Cys Leu Arg Asn Gly Ile
420 425 430
Pro Lys Gly Ala Ala Ile Leu Val Ala Phe Phe Met Ser Ala Val Phe
435 440 445
His Glu Leu Cys Ile Ala Val Pro Cys His Ile Phe Lys Phe Trp Ala
450 455 460
Phe Ile Gly Ile Met Phe Gln Val Pro Leu Val Leu Leu Thr Asn Tyr
465 470 475 480
Leu Gln His Lys Phe Gln Asn Ser Met Val Gly Asn Met Ile Phe Trp
485 490 495
Cys Phe Phe Ser Ile Phe Gly Gln Pro Met Cys Val Leu Leu Tyr Tyr
500 505 510
His Asp Val Met Asn Gln Lys Gly Lys Ser Lys
515 520
<210> 84
<211> 517
<212> БЕЛОК
<213> Vernonia galamensis
<400> 84
Met Ala Leu Leu Asp Thr Pro Gln Ile Gly Glu Ile Thr Thr Thr Ala
1 5 10 15
Thr Thr Thr Ile Arg Gln His Pro Leu Gly Lys Pro Asp Ala Gly Ile
20 25 30
Gly Asp Gly Leu Phe Ser Ser Ser Ser Ser Lys Thr Asn Ser Ser Phe
35 40 45
Glu Asp Gly Asp Ser Leu Asn Gly Asp Phe Asn Asp Lys Phe Lys Glu
50 55 60
Gln Ile Gly Ala Gly Asp Glu Ser Lys Lys Gly Asn Gly Lys Ile Asp
65 70 75 80
His Gly Gly Val Lys Lys Gly Arg Glu Thr Thr Val Val His Tyr Ala
85 90 95
Tyr Arg Pro Ser Ser Pro Ala His Arg Arg Ile Lys Glu Ser Pro Leu
100 105 110
Ser Ser Asp Ala Ile Phe Lys Gln Ser His Ala Gly Leu Phe Asn Leu
115 120 125
Cys Ile Val Val Leu Val Ala Val Asn Gly Arg Leu Ile Ile Glu Asn
130 135 140
Leu Met Lys Tyr Gly Leu Leu Ile Asn Ser Lys Phe Trp Phe Ser Ser
145 150 155 160
Arg Ser Leu Arg Asp Trp Pro Leu Leu Met Cys Trp Leu Thr Pro Ser
165 170 175
Asp Phe Pro Leu Ala Ala Tyr Ile Val Glu Lys Leu Ala Trp Lys Lys
180 185 190
Arg Ile Ser Asp Pro Val Val Ile Thr Leu His Val Val Ile Thr Thr
195 200 205
Thr Ala Ile Leu Tyr Pro Ile Phe Met Ile Leu Arg Phe Asp Ser Val
210 215 220
Val Leu Leu Gly Val Ser Leu Met Leu Cys Ala Cys Ile Asn Trp Leu
225 230 235 240
Lys Leu Val Ser Phe Val His Thr Asn Tyr Asp Met Arg Ser Leu Leu
245 250 255
Asn Ser Thr Gly Lys Gly Glu Val Glu Pro Met Ser Ser Asn Met Asp
260 265 270
Tyr Phe Tyr Asp Ile Asn Phe Lys Ser Leu Val Tyr Phe Met Val Ala
275 280 285
Pro Thr Leu Cys Tyr Gln Ile Ser Tyr Pro Arg Thr Ala Phe Ile Arg
290 295 300
Lys Gly Trp Val Phe Arg Gln Leu Ile Lys Leu Val Ile Phe Thr Gly
305 310 315 320
Phe Met Gly Phe Ile Ile Glu Gln Tyr Ile Asn Pro Ile Val Lys Asn
325 330 335
Ser Arg His Pro Leu Asn Gly Asp Phe Leu Tyr Ala Ile Glu Arg Val
340 345 350
Leu Lys Val Ser Val Pro Asn Leu Tyr Val Trp Leu Cys Met Phe Tyr
355 360 365
Cys Phe Phe His Leu Trp Leu Asn Ile Leu Ala Glu Leu Leu Trp Phe
370 375 380
Gly Asp Arg Glu Phe Tyr Lys Asp Trp Trp Asn Thr Gln Thr Ile Glu
385 390 395 400
Glu Tyr Trp Arg Leu Trp Asn Met Pro Val His Lys Trp Ile Val Arg
405 410 415
His Leu Tyr Phe Pro Cys Leu Arg Asn Gly Ile Ser Lys Gly Ala Ala
420 425 430
Ile Leu Val Ala Phe Phe Met Ser Ala Val Phe His Glu Leu Cys Ile
435 440 445
Ala Val Pro Cys His Ile Leu Lys Phe Trp Ala Phe Ile Gly Ile Met
450 455 460
Phe Gln Val Pro Leu Val Leu Leu Thr Asn Tyr Leu Gln His Lys Phe
465 470 475 480
Gln Asn Ser Met Val Gly Asn Met Ile Phe Trp Cys Phe Phe Ser Ile
485 490 495
Phe Gly Gln Pro Met Cys Val Phe Leu Tyr Tyr His Glu Val Asn Gln
500 505 510
Lys Gly Lys Ser Lys
515
<210> 85
<211> 507
<212> БЕЛОК
<213> Euonymus alatus
<400> 85
Met Ala Ala Asn Leu Asn Glu Ala Ser Asp Leu Asn Phe Ser Leu Arg
1 5 10 15
Arg Arg Thr Gly Gly Ile Ser Ser Thr Thr Val Pro Asp Ser Ser Ser
20 25 30
Glu Thr Ser Ser Ser Glu Ala Asp Tyr Leu Asp Gly Gly Lys Gly Ala
35 40 45
Ala Asp Val Lys Asp Arg Gly Asp Gly Ala Val Glu Phe Gln Asn Ser
50 55 60
Met Lys Asn Val Glu Arg Ile Glu Lys His Glu Ser Arg Val Gly Leu
65 70 75 80
Asp Ser Arg Phe Thr Tyr Arg Pro Ser Val Pro Ala His Arg Thr Ile
85 90 95
Lys Glu Ser Pro Leu Ser Ser Asp Ala Ile Phe Lys Gln Ser His Ala
100 105 110
Gly Leu Phe Asn Leu Cys Ile Val Val Leu Val Ala Val Asn Ser Arg
115 120 125
Leu Ile Ile Glu Asn Leu Met Lys Tyr Gly Trp Leu Ile Arg Ser Gly
130 135 140
Phe Trp Phe Ser Ser Arg Ser Leu Arg Asp Trp Pro Leu Phe Met Cys
145 150 155 160
Cys Leu Thr Leu Pro Val Phe Pro Leu Ala Ala Phe Leu Phe Glu Lys
165 170 175
Leu Ala Gln Lys Asn Leu Ile Ser Glu Pro Val Val Val Leu Leu His
180 185 190
Ile Val Asn Thr Thr Ala Ala Val Leu Tyr Pro Val Leu Val Ile Leu
195 200 205
Arg Cys Asp Ser Ala Phe Met Ser Gly Val Thr Leu Met Leu Phe Ala
210 215 220
Cys Ile Val Trp Leu Lys Leu Val Ser Tyr Ala His Thr Asn Tyr Asp
225 230 235 240
Met Arg Ala Leu Thr Lys Ser Val Glu Lys Gly Asp Thr Pro Leu Ser
245 250 255
Ser Gln Asn Met Asp Tyr Ser Phe Asp Val Asn Ile Lys Ser Leu Ala
260 265 270
Tyr Phe Met Val Ala Pro Thr Leu Cys Tyr Gln Ile Ser Tyr Pro Arg
275 280 285
Thr Pro Tyr Val Arg Lys Gly Trp Val Val Arg Gln Phe Val Lys Leu
290 295 300
Ile Ile Phe Thr Gly Leu Met Gly Phe Ile Ile Glu Gln Tyr Ile Asn
305 310 315 320
Pro Ile Val Gln Asn Ser Gln His Pro Leu Lys Gly Asn Phe Leu Tyr
325 330 335
Ala Ile Glu Arg Val Leu Lys Leu Ser Val Pro Asn Leu Tyr Val Trp
340 345 350
Leu Cys Met Phe Tyr Cys Leu Phe His Leu Trp Leu Asn Ile Leu Ala
355 360 365
Glu Leu Leu Cys Phe Gly Asp Arg Glu Phe Tyr Lys Asp Trp Trp Asn
370 375 380
Ala Lys Thr Val Glu Glu Tyr Trp Arg Met Trp Asn Met Pro Val His
385 390 395 400
Lys Trp Met Val Arg His Ile Tyr Phe Pro Cys Leu Arg Asn Gly Ile
405 410 415
Pro Lys Gly Val Ala Phe Val Ile Ser Phe Leu Val Ser Ala Val Phe
420 425 430
His Glu Leu Cys Ile Ala Val Pro Cys His Ile Phe Lys Leu Trp Ala
435 440 445
Phe Phe Gly Ile Met Leu Gln Val Pro Leu Val Leu Ile Thr Ser Tyr
450 455 460
Leu Gln Asn Lys Phe Arg Ser Ser Met Val Gly Asn Met Met Phe Trp
465 470 475 480
Phe Ser Phe Cys Ile Phe Gly Gln Pro Met Cys Leu Leu Leu Tyr Tyr
485 490 495
His Asp Leu Met Asn Arg Asn Gly Lys Met Glu
500 505
<210> 86
<211> 498
<212> БЕЛОК
<213> Caenorhabditis elegans
<400> 86
Met Gln Met Arg Gln Gln Thr Gly Arg Arg Arg Arg Gln Pro Ser Glu
1 5 10 15
Thr Ser Asn Gly Ser Leu Ala Ser Ser Arg Arg Ser Ser Phe Ala Gln
20 25 30
Asn Gly Asn Ser Ser Arg Lys Ser Ser Glu Met Arg Gly Pro Cys Glu
35 40 45
Lys Val Val His Thr Ala Gln Asp Ser Leu Phe Ser Thr Ser Ser Gly
50 55 60
Trp Thr Asn Phe Arg Gly Phe Phe Asn Leu Ser Ile Leu Leu Leu Val
65 70 75 80
Leu Ser Asn Gly Arg Val Ala Leu Glu Asn Val Ile Lys Tyr Gly Ile
85 90 95
Leu Ile Thr Pro Leu Gln Trp Ile Ser Thr Phe Val Glu His His Tyr
100 105 110
Ser Ile Trp Ser Trp Pro Asn Leu Ala Leu Ile Leu Cys Ser Asn Ile
115 120 125
Gln Ile Leu Ser Val Phe Gly Met Glu Lys Ile Leu Glu Arg Gly Trp
130 135 140
Leu Gly Asn Gly Phe Ala Ala Val Phe Tyr Thr Ser Leu Val Ile Ala
145 150 155 160
His Leu Thr Ile Pro Val Val Val Thr Leu Thr His Lys Trp Lys Asn
165 170 175
Pro Leu Trp Ser Val Val Met Met Gly Val Tyr Val Ile Glu Ala Leu
180 185 190
Lys Phe Ile Ser Tyr Gly His Val Asn Tyr Trp Ala Arg Asp Ala Arg
195 200 205
Arg Lys Ile Thr Glu Leu Lys Thr Gln Val Thr Asp Leu Ala Lys Lys
210 215 220
Thr Cys Asp Pro Lys Gln Phe Trp Asp Leu Lys Asp Glu Leu Ser Met
225 230 235 240
His Gln Met Ala Ala Gln Tyr Pro Ala Asn Leu Thr Leu Ser Asn Ile
245 250 255
Tyr Tyr Phe Met Ala Ala Pro Thr Leu Cys Tyr Glu Phe Lys Phe Pro
260 265 270
Arg Leu Leu Arg Ile Arg Lys His Phe Leu Ile Lys Arg Thr Val Glu
275 280 285
Leu Ile Phe Leu Ser Phe Leu Ile Ala Ala Leu Val Gln Gln Trp Val
290 295 300
Val Pro Thr Val Arg Asn Ser Met Lys Pro Leu Ser Glu Met Glu Tyr
305 310 315 320
Ser Arg Cys Leu Glu Arg Leu Leu Lys Leu Ala Ile Pro Asn His Leu
325 330 335
Ile Trp Leu Leu Phe Phe Tyr Thr Phe Phe His Ser Phe Leu Asn Leu
340 345 350
Ile Ala Glu Leu Leu Arg Phe Ala Asp Arg Glu Phe Tyr Arg Asp Phe
355 360 365
Trp Asn Ala Glu Thr Ile Gly Tyr Phe Trp Lys Ser Trp Asn Ile Pro
370 375 380
Val His Arg Phe Ala Val Arg His Ile Tyr Ser Pro Met Met Arg Asn
385 390 395 400
Asn Phe Ser Lys Met Ser Ala Phe Phe Val Val Phe Phe Val Ser Ala
405 410 415
Phe Phe His Glu Tyr Leu Val Ser Val Pro Leu Lys Ile Phe Arg Leu
420 425 430
Trp Ser Tyr Tyr Gly Met Met Gly Gln Ile Pro Leu Ser Ile Ile Thr
435 440 445
Asp Lys Val Val Arg Gly Gly Arg Thr Gly Asn Ile Ile Val Trp Leu
450 455 460
Ser Leu Ile Val Gly Gln Pro Leu Ala Ile Leu Met Tyr Gly His Asp
465 470 475 480
Trp Tyr Ile Leu Asn Phe Gly Val Ser Ala Val Gln Asn Gln Thr Val
485 490 495
Gly Ile
<210> 87
<211> 498
<212> БЕЛОК
<213> Rattus norvegicus
<400> 87
Met Gly Asp Arg Gly Gly Ala Gly Ser Ser Arg Arg Arg Arg Thr Gly
1 5 10 15
Ser Arg Val Ser Val Gln Gly Gly Ser Gly Pro Lys Val Glu Glu Asp
20 25 30
Glu Val Arg Glu Ala Ala Val Ser Pro Asp Leu Gly Ala Gly Gly Asp
35 40 45
Ala Pro Ala Pro Ala Pro Ala Pro Ala His Thr Arg Asp Lys Asp Arg
50 55 60
Gln Thr Ser Val Gly Asp Gly His Trp Glu Leu Arg Cys His Arg Leu
65 70 75 80
Gln Asp Ser Leu Phe Ser Ser Asp Ser Gly Phe Ser Asn Tyr Arg Gly
85 90 95
Ile Leu Asn Trp Cys Val Val Met Leu Ile Leu Ser Asn Ala Arg Leu
100 105 110
Ser Leu Glu Asn Leu Ile Lys Tyr Gly Ile Leu Val Asp Pro Ile Gln
115 120 125
Val Val Ser Leu Phe Leu Lys Asp Pro Tyr Ser Trp Pro Ala Pro Cys
130 135 140
Leu Ile Ile Ala Ser Asn Ile Phe Ile Val Ala Thr Phe Gln Ile Glu
145 150 155 160
Lys Arg Leu Ser Val Gly Ala Leu Thr Glu Gln Met Gly Leu Leu Leu
165 170 175
His Val Val Asn Leu Ala Thr Ile Ile Cys Phe Pro Ala Ala Val Ala
180 185 190
Leu Leu Val Glu Ser Ile Thr Pro Val Gly Ser Leu Phe Ala Leu Ala
195 200 205
Ser Tyr Ser Ile Ile Phe Leu Lys Leu Ser Ser Tyr Arg Asp Val Asn
210 215 220
Leu Trp Cys Arg Gln Arg Arg Val Lys Ala Lys Ala Val Ser Ala Gly
225 230 235 240
Lys Lys Val Ser Gly Ala Ala Ala Gln Asn Thr Val Ser Tyr Pro Asp
245 250 255
Asn Leu Thr Tyr Arg Asp Leu Tyr Tyr Phe Ile Phe Ala Pro Thr Leu
260 265 270
Cys Tyr Glu Leu Asn Phe Pro Arg Ser Pro Arg Ile Arg Lys Arg Phe
275 280 285
Leu Leu Arg Arg Val Leu Glu Met Leu Phe Phe Thr Gln Leu Gln Val
290 295 300
Gly Leu Ile Gln Gln Trp Met Val Pro Thr Ile Gln Asn Ser Met Lys
305 310 315 320
Pro Phe Lys Asp Met Asp Tyr Ser Arg Ile Ile Glu Arg Leu Leu Lys
325 330 335
Leu Ala Val Pro Asn His Leu Ile Trp Leu Ile Phe Phe Tyr Trp Leu
340 345 350
Phe His Ser Cys Leu Asn Ala Val Ala Glu Leu Leu Gln Phe Gly Asp
355 360 365
Arg Glu Phe Tyr Arg Asp Trp Trp Asn Ala Glu Ser Val Thr Tyr Phe
370 375 380
Trp Gln Asn Trp Asn Ile Pro Val His Lys Trp Cys Ile Arg His Phe
385 390 395 400
Tyr Lys Pro Met Leu Arg Leu Gly Ser Asn Lys Trp Met Ala Arg Thr
405 410 415
Gly Val Phe Trp Ala Ser Ala Phe Phe His Glu Tyr Leu Val Ser Ile
420 425 430
Pro Leu Arg Met Phe Arg Leu Trp Ala Phe Thr Ala Met Met Ala Gln
435 440 445
Val Pro Leu Ala Trp Ile Val Asn Arg Phe Phe Gln Gly Asn Tyr Gly
450 455 460
Asn Ala Ala Val Trp Val Thr Leu Ile Ile Gly Gln Pro Val Ala Val
465 470 475 480
Leu Met Tyr Val His Asp Tyr Tyr Val Leu Asn Tyr Asp Ala Pro Val
485 490 495
Gly Ala
<210> 88
<211> 488
<212> БЕЛОК
<213> Homo sapiens
<400> 88
Met Gly Asp Arg Gly Ser Ser Arg Arg Arg Arg Thr Gly Ser Arg Pro
1 5 10 15
Ser Ser His Gly Gly Gly Gly Pro Ala Ala Ala Glu Glu Glu Val Arg
20 25 30
Asp Ala Ala Ala Gly Pro Asp Val Gly Ala Ala Gly Asp Ala Pro Ala
35 40 45
Pro Ala Pro Asn Lys Asp Gly Asp Ala Gly Val Gly Ser Gly His Trp
50 55 60
Glu Leu Arg Cys His Arg Leu Gln Asp Ser Leu Phe Ser Ser Asp Ser
65 70 75 80
Gly Phe Ser Asn Tyr Arg Gly Ile Leu Asn Trp Cys Val Val Met Leu
85 90 95
Ile Leu Ser Asn Ala Arg Leu Phe Leu Glu Asn Leu Ile Lys Tyr Gly
100 105 110
Ile Leu Val Asp Pro Ile Gln Val Val Ser Leu Phe Leu Lys Asp Pro
115 120 125
Tyr Ser Trp Pro Ala Pro Cys Leu Val Ile Ala Ala Asn Val Phe Ala
130 135 140
Val Ala Ala Phe Gln Val Glu Lys Arg Leu Ala Val Gly Ala Leu Thr
145 150 155 160
Glu Gln Ala Gly Leu Leu Leu His Val Ala Asn Leu Ala Thr Ile Leu
165 170 175
Cys Phe Pro Ala Ala Val Val Leu Leu Val Glu Ser Ile Thr Pro Val
180 185 190
Gly Ser Leu Leu Ala Leu Met Ala His Thr Ile Leu Phe Leu Lys Leu
195 200 205
Phe Ser Tyr Arg Asp Val Asn Ser Trp Cys Arg Arg Ala Arg Ala Lys
210 215 220
Ala Ala Ser Ala Gly Lys Lys Ala Ser Ser Ala Ala Ala Pro His Thr
225 230 235 240
Val Ser Tyr Pro Asp Asn Leu Thr Tyr Arg Asp Leu Tyr Tyr Phe Leu
245 250 255
Phe Ala Pro Thr Leu Cys Tyr Glu Leu Asn Phe Pro Arg Ser Pro Arg
260 265 270
Ile Arg Lys Arg Phe Leu Leu Arg Arg Ile Leu Glu Met Leu Phe Phe
275 280 285
Thr Gln Leu Gln Val Gly Leu Ile Gln Gln Trp Met Val Pro Thr Ile
290 295 300
Gln Asn Ser Met Lys Pro Phe Lys Asp Met Asp Tyr Ser Arg Ile Ile
305 310 315 320
Glu Arg Leu Leu Lys Leu Ala Val Pro Asn His Leu Ile Trp Leu Ile
325 330 335
Phe Phe Tyr Trp Leu Phe His Ser Cys Leu Asn Ala Val Ala Glu Leu
340 345 350
Met Gln Phe Gly Asp Arg Glu Phe Tyr Arg Asp Trp Trp Asn Ser Glu
355 360 365
Ser Val Thr Tyr Phe Trp Gln Asn Trp Asn Ile Pro Val His Lys Trp
370 375 380
Cys Ile Arg His Phe Tyr Lys Pro Met Leu Arg Arg Gly Ser Ser Lys
385 390 395 400
Trp Met Ala Arg Thr Gly Val Phe Leu Ala Ser Ala Phe Phe His Glu
405 410 415
Tyr Leu Val Ser Val Pro Leu Arg Met Phe Arg Leu Trp Ala Phe Thr
420 425 430
Gly Met Met Ala Gln Ile Pro Leu Ala Trp Phe Val Gly Arg Phe Phe
435 440 445
Gln Gly Asn Tyr Gly Asn Ala Ala Val Trp Leu Ser Leu Ile Ile Gly
450 455 460
Gln Pro Ile Ala Val Leu Met Tyr Val His Asp Tyr Tyr Val Leu Asn
465 470 475 480
Tyr Glu Ala Pro Ala Ala Glu Ala
485
<210> 89
<211> 11
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> консервативная последовательность
<220>
<221> X
<222> (4)..(4)
<223> треонин (T) или серин (S)
<400> 89
Arg Gly Val Xaa Arg His Arg Trp Thr Gly Arg
1 5 10
<210> 90
<211> 8
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> консервативная последовательность
<220>
<221> X
<222> (1)..(1)
<223> фенилаланин (F) или тирозин (Y)
<400> 90
Xaa Glu Ala His Leu Trp Asp Lys
1 5
<210> 91
<211> 9
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> консервативная последовательность
<400> 91
Asp Leu Ala Ala Leu Lys Tyr Trp Gly
1 5
<210> 92
<211> 8
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> консервативная последовательность
<220>
<221> неопределенный признак
<222> (2)..(2)
<223> Xaa может представлять собой любую природную аминокислоту
<220>
<221> X
<222> (5)..(5)
<223> серин (S) или аланин (A)
<220>
<221> X
<222> (8)..(8)
<223> любая аминокислота
<400> 92
Ser Xaa Gly Phe Xaa Arg Gly Xaa
1 5
<210> 93
<211> 14
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> консервативная последовательность
<220>
<221> X
<222> (3)..(3)
<223> гистидин (H) или глутамин (Q)
<220>
<221> X
<222> (6)..(6)
<223> аргинин (R) или лизин (K)
<220>
<221> X
<222> (12)..(12)
<223> аргинин (R) или лизин (K)
<220>
<221> неопределенный признак
<222> (13)..(13)
<223> Xaa может представлять собой любую природную аминокислоту
<400> 93
His His Xaa Asn Gly Xaa Trp Glu Ala Arg Ile Gly Xaa Val
1 5 10
<210> 94
<211> 9
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> консервативная последовательность
<220>
<221> X
<222> (7)..(7)
<223> любая аминокислота
<400> 94
Gln Glu Glu Ala Ala Ala Xaa Tyr Asp
1 5
<210> 95
<211> 165
<212> БЕЛОК
<213> Brassica napus
<400> 95
Met Gly Ile Leu Arg Lys Lys Lys His Glu Arg Lys Pro Ser Phe Lys
1 5 10 15
Ser Val Leu Thr Ala Ile Leu Ala Thr His Ala Ala Thr Phe Leu Leu
20 25 30
Leu Ile Ala Gly Val Ser Leu Ala Gly Thr Ala Ala Ala Phe Ile Ala
35 40 45
Thr Met Pro Leu Phe Val Val Phe Ser Pro Ile Leu Val Pro Ala Gly
50 55 60
Ile Thr Thr Gly Leu Leu Thr Thr Gly Leu Ala Ala Ala Gly Gly Ala
65 70 75 80
Gly Ala Thr Ala Val Thr Ile Ile Leu Trp Leu Tyr Lys Arg Ala Thr
85 90 95
Gly Lys Ala Pro Pro Lys Val Leu Glu Lys Val Leu Lys Lys Ile Ile
100 105 110
Pro Gly Ala Ala Ala Ala Pro Ala Ala Ala Pro Gly Ala Ala Pro Ala
115 120 125
Ala Ala Pro Ala Ala Ala Pro Ala Val Ala Pro Ala Ala Ala Pro Ala
130 135 140
Ala Ala Pro Ala Pro Lys Pro Ala Ala Pro Pro Ala Pro Lys Pro Ala
145 150 155 160
Ala Ala Pro Ser Ile
165
<210> 96
<211> 193
<212> БЕЛОК
<213> Brassica napus
<400> 96
Met Ala Asp Val Arg Thr His Ala His Gln Val Gln Val His Pro Leu
1 5 10 15
Arg Gln Gln Glu Gly Gly Ile Lys Val Val Tyr Pro Gln Ser Gly Pro
20 25 30
Ser Ser Thr Gln Val Leu Ala Val Ile Ala Gly Val Pro Val Gly Gly
35 40 45
Thr Leu Leu Thr Leu Ala Gly Leu Thr Leu Ala Gly Ser Val Ile Gly
50 55 60
Leu Met Leu Ala Phe Pro Leu Phe Leu Ile Phe Ser Pro Val Ile Val
65 70 75 80
Pro Ala Ala Phe Val Ile Gly Leu Ala Met Thr Gly Phe Met Ala Ser
85 90 95
Gly Ala Ile Gly Leu Thr Gly Leu Ser Ser Met Ser Trp Val Leu Asn
100 105 110
His Ile Arg Arg Val Arg Glu Arg Met Pro Asp Glu Leu Glu Glu Ala
115 120 125
Lys Gln Arg Leu Ala Asp Met Ala Glu Tyr Val Gly Gln Arg Thr Lys
130 135 140
Asp Ala Gly Gln Thr Ile Glu Glu Lys Ala His Asp Val Arg Glu Ser
145 150 155 160
Lys Thr Tyr Asp Val Arg Asp Arg Asp Thr Lys Gly His Thr Ala Thr
165 170 175
Gly Gly Asp Arg Asp Thr Lys Thr Thr Arg Glu Val Arg Val Ala Thr
180 185 190
Thr
<210> 97
<211> 188
<212> БЕЛОК
<213> Brassica napus
<400> 97
Met Ala Asn Val Asp Arg Arg Val Asn Val Asp Arg Thr Asp Lys Gly
1 5 10 15
Leu Gln Leu Gln Pro Gln Tyr Glu Asp Arg Val Gly Tyr Gly Tyr Gly
20 25 30
Tyr Gly Gly Asn Thr Asp Tyr Lys Ser Cys Gly Pro Ser Thr Asn Gln
35 40 45
Ile Val Ala Leu Ile Ala Gly Val Pro Ile Gly Gly Ser Leu Leu Ala
50 55 60
Leu Ala Gly Leu Thr Leu Ala Gly Ser Val Ile Gly Phe Met Leu Ser
65 70 75 80
Ile Pro Leu Phe Leu Leu Phe Ser Pro Val Ile Val Pro Ala Ala Leu
85 90 95
Thr Ile Gly Leu Ala Val Thr Gly Ile Leu Ala Ser Gly Leu Phe Gly
100 105 110
Leu Thr Gly Leu Ser Ser Val Ser Trp Val Leu Asn Tyr Ile Arg Gly
115 120 125
Arg Ser Asp Thr Val Pro Glu Gln Leu Asp Tyr Ala Lys Arg Arg Met
130 135 140
Ala Asp Ala Val Gly Tyr Ala Gly Gln Lys Gly Lys Glu Met Gly Gln
145 150 155 160
Tyr Val Gln Asp Lys Ala His Glu Ala His Asp Thr Ser Leu Thr Thr
165 170 175
Glu Thr Asn Gly Lys Thr Arg Arg Ala His Ile Ala
180 185
<210> 98
<211> 180
<212> БЕЛОК
<213> Brassica napus
<400> 98
Met Ala Asp Thr Ala Arg Thr His His Asp Ile Thr Ser Arg Asp Gln
1 5 10 15
Tyr Pro Ile Leu Gly Arg Asp Arg Asp Gln Tyr Pro Tyr Gly Arg Ser
20 25 30
Asp Tyr Gln Thr Ser Gly Gln Asp Tyr Ser Lys Thr Arg Gln Ile Ala
35 40 45
Lys Ala Ala Thr Ala Val Thr Ala Gly Gly Ser Leu Leu Val Leu Ser
50 55 60
Ser Leu Thr Leu Val Gly Thr Val Ile Ala Leu Thr Val Ala Thr Thr
65 70 75 80
Leu Leu Val Ile Phe Ser Pro Ile Leu Val Pro Ala Leu Ile Thr Val
85 90 95
Ala Leu Leu Ile Thr Gly Phe Leu Ser Ser Gly Gly Phe Gly Ile Ala
100 105 110
Asp Ile Thr Val Phe Ser Trp Ile Tyr Lys Tyr Ala Thr Gly Glu His
115 120 125
Pro Gln Gly Ser Asp Lys Leu Asp Ser Ala Arg Met Lys Leu Gly Thr
130 135 140
Lys Ala Gln Asp Ile Lys Asp Arg Ala Gln Tyr Tyr Gly Gln Gln His
145 150 155 160
Thr Gly Gly Glu His Asp Arg Asp Arg Thr Arg Gly Thr His His Thr
165 170 175
Thr Thr Thr Thr
180
<210> 99
<211> 210
<212> БЕЛОК
<213> Brassica napus
<400> 99
Met Ala Asp Thr His Arg Val Asp Arg Thr Asp Arg His Leu Gln Phe
1 5 10 15
Gln Ser Pro Tyr Glu Gly Gly Arg Val Ser Ile Gln Tyr Glu Gly Gly
20 25 30
Gly Gly Ala Gly Gly Tyr Gly Gly Arg Gly Gly Gly Tyr Gly Ala Glu
35 40 45
Gly Tyr Lys Ser Met Met Pro Glu Arg Gly Pro Ser Ser Thr Gln Val
50 55 60
Leu Ser Phe Leu Val Gly Val Pro Ile Val Gly Ser Leu Leu Ala Ile
65 70 75 80
Ala Gly Leu Leu Leu Ala Gly Ser Val Ile Gly Leu Leu Ile Ser Ile
85 90 95
Pro Leu Phe Leu Leu Phe Ser Pro Val Ile Val Pro Ala Ala Leu Thr
100 105 110
Ile Gly Leu Ala Ala Thr Gly Phe Leu Ala Ser Gly Met Phe Gly Leu
115 120 125
Thr Gly Leu Ser Ser Val Ser Trp Val Leu Asn Tyr Leu Arg Gly Thr
130 135 140
Arg Lys Ser Ser Val Pro Glu Gln Leu Glu Tyr Ala Lys Lys Arg Met
145 150 155 160
Ala Asp Ala Val Gly Tyr Ala Gly Gln Lys Gly Lys Gly Met Gly Gln
165 170 175
His Val Gln Asn Lys Ala Gln Glu Ala Lys Gln Tyr Asp Ile Ser Lys
180 185 190
Thr His Asp Thr Thr Thr Lys Gly His Glu Thr Thr Gln Arg Thr Ala
195 200 205
Ala Ala
210
<210> 100
<211> 149
<212> БЕЛОК
<213> Brassica napus
<400> 100
Met Ala Asn Gln Thr Arg Thr His Gln Asp Ile Ile Val Arg Asp Ser
1 5 10 15
Arg Ile Thr Leu Asp Arg Asp His Pro Lys Thr Gly Ala Gln Met Val
20 25 30
Lys Val Ala Thr Gly Val Ala Ala Gly Gly Ser Leu Leu Val Leu Ser
35 40 45
Gly Leu Thr Leu Ala Gly Thr Val Ile Ala Phe Ala Val Ala Thr Pro
50 55 60
Leu Leu Ile Ile Phe Ser Pro Val Leu Val Pro Ala Val Ile Thr Val
65 70 75 80
Val Leu Ile Ile Thr Gly Phe Leu Ala Ser Gly Gly Phe Gly Ile Ala
85 90 95
Ala Ile Thr Ala Phe Ser Trp Leu Tyr Arg His Met Thr Gly Ser Gly
100 105 110
Ser Asp Gln Lys Ile Glu Ser Ala Arg Met Lys Val Gly Ser Arg Gly
115 120 125
Tyr Asp Thr Lys Tyr Gly Gln His Asn Ile Gly Val His Gln Gln His
130 135 140
Gln Gln Ala Ala Ser
145
<210> 101
<211> 137
<212> БЕЛОК
<213> Arachis hypogaea
<400> 101
Met Ala Glu Ala Leu Tyr Tyr Gly Gly Arg Gln Arg Gln Glu Gln Pro
1 5 10 15
Arg Ser Thr Gln Leu Val Lys Ala Thr Thr Ala Val Val Ala Gly Gly
20 25 30
Ser Leu Leu Ile Leu Ala Gly Leu Val Leu Ala Gly Thr Val Ile Gly
35 40 45
Leu Thr Thr Ile Thr Pro Leu Phe Val Ile Phe Ser Pro Val Leu Val
50 55 60
Pro Ala Val Ile Thr Val Ala Leu Leu Gly Leu Gly Phe Leu Ala Ser
65 70 75 80
Gly Gly Phe Gly Val Ala Ala Ile Thr Val Leu Thr Trp Ile Tyr Arg
85 90 95
Tyr Val Thr Gly Lys His Pro Pro Gly Ala Asn Gln Leu Asp Thr Ala
100 105 110
Arg His Lys Leu Met Gly Lys Ala Arg Glu Ile Lys Asp Phe Gly Gln
115 120 125
Gln Gln Thr Ser Gly Ala Gln Ala Ser
130 135
<210> 102
<211> 150
<212> БЕЛОК
<213> Arachis hypogaea
<400> 102
Met Thr Asp Arg Thr Gln Pro His Ala Val Gln Val His Thr Thr Ala
1 5 10 15
Gly Arg Phe Gly Asp Thr Ala Ala Gly Thr Asn Arg Tyr Ala Asp Arg
20 25 30
Gly Pro Ser Thr Ser Lys Val Ile Ala Val Ile Thr Gly Leu Pro Ile
35 40 45
Gly Gly Thr Leu Leu Leu Phe Ala Gly Leu Ala Leu Ala Gly Thr Leu
50 55 60
Leu Gly Leu Ala Val Thr Thr Pro Leu Phe Ile Leu Phe Ser Pro Val
65 70 75 80
Ile Val Pro Ala Thr Ile Val Val Gly Leu Ser Val Ala Gly Phe Leu
85 90 95
Thr Ser Gly Ala Cys Gly Leu Thr Gly Leu Ser Ser Phe Ser Trp Val
100 105 110
Met Asn Tyr Ile Arg Gln Thr His Gly Ser Val Pro Glu Gln Leu Glu
115 120 125
Met Ala Lys His Arg Met Ala Asp Val Ala Gly Tyr Val Gly Gln Lys
130 135 140
Thr Lys Asp Val Gly Gln
145 150
<210> 103
<211> 166
<212> БЕЛОК
<213> Arachis hypogaea
<400> 103
Met Ser Asp Gln Thr Arg Thr Gly Tyr Gly Gly Gly Gly Ser Tyr Gly
1 5 10 15
Ser Ser Tyr Gly Gly Gly Gly Thr Tyr Gly Ser Ser Tyr Gly Thr Ser
20 25 30
Tyr Asp Pro Ser Thr Asn Gln Pro Ile Arg Gln Ala Ile Lys Phe Met
35 40 45
Thr Ala Ser Thr Ile Gly Val Ser Phe Leu Ile Leu Ser Gly Leu Ile
50 55 60
Leu Thr Gly Thr Val Ile Gly Leu Ile Ile Ala Thr Pro Leu Leu Val
65 70 75 80
Ile Phe Ser Pro Ile Leu Val Pro Ala Ala Ile Thr Leu Ala Leu Ala
85 90 95
Ala Gly Gly Phe Leu Phe Ser Gly Gly Cys Gly Val Ala Ala Ile Ala
100 105 110
Ala Leu Ser Trp Leu Tyr Ser Tyr Val Thr Gly Lys His Pro Ala Gly
115 120 125
Ser Asp Arg Leu Asp Tyr Ala Lys Gly Val Ile Ala Asp Lys Ala Arg
130 135 140
Asp Val Lys Asp Arg Ala Lys Asp Tyr Ala Gly Ala Gly Arg Ala Gln
145 150 155 160
Glu Gly Thr Pro Gly Tyr
165
<210> 104
<211> 176
<212> БЕЛОК
<213> Arachis hypogaea
<400> 104
Met Ala Thr Ala Thr Asp Arg Ala Pro His Gln Val Gln Val His Thr
1 5 10 15
Pro Thr Thr Gln Arg Val Asp Val Pro Arg Arg Gly Tyr Asp Val Ser
20 25 30
Gly Gly Gly Ile Lys Thr Leu Leu Pro Glu Arg Gly Pro Ser Thr Ser
35 40 45
Gln Ile Ile Ala Val Leu Val Gly Val Pro Thr Gly Gly Thr Leu Leu
50 55 60
Leu Leu Ser Gly Leu Ser Leu Leu Gly Thr Ile Ile Gly Leu Ala Ile
65 70 75 80
Ala Thr Pro Val Phe Ile Phe Phe Ser Pro Val Ile Val Pro Ala Val
85 90 95
Val Thr Ile Gly Leu Ala Val Thr Gly Ile Leu Thr Ala Gly Ala Cys
100 105 110
Gly Leu Thr Gly Leu Met Ser Leu Ser Trp Met Ile Asn Phe Ile Arg
115 120 125
Gln Val His Gly Thr Thr Val Pro Asp Gln Leu Asp Ser Val Lys Arg
130 135 140
Arg Met Ala Asp Met Ala Asp Tyr Val Gly Gln Lys Thr Lys Asp Ala
145 150 155 160
Gly Gln Glu Ile Gln Thr Lys Ala Gln Asp Val Lys Arg Ser Ser Ser
165 170 175
<210> 105
<211> 153
<212> БЕЛОК
<213> Ricinus communis
<400> 105
Met Ala Asp Arg Pro Gln Pro His Gln Val Gln Val His Arg Tyr Asp
1 5 10 15
Pro Thr Thr Gly Tyr Lys Gly Gln Gln Lys Gly Pro Ser Ala Ser Lys
20 25 30
Val Leu Ala Val Leu Thr Phe Leu Pro Val Gly Gly Gly Leu Leu Ser
35 40 45
Leu Ser Gly Ile Thr Leu Thr Asn Thr Leu Ile Gly Met Ala Ile Ala
50 55 60
Thr Pro Leu Phe Ile Leu Phe Gly Pro Ile Ile Leu Pro Ala Ala Val
65 70 75 80
Val Ile Gly Leu Ala Met Met Ala Phe Met Val Ala Gly Ala Leu Gly
85 90 95
Leu Ser Gly Leu Thr Ser Gln Ser Trp Ala Leu Lys Tyr Phe Arg Glu
100 105 110
Gly Thr Ala Met Pro Glu Ser Leu Asp Gln Ala Lys Lys Arg Met Gln
115 120 125
Asp Met Ala Gly Tyr Val Gly Met Lys Thr Lys Glu Val Gly Gln Asp
130 135 140
Ile Gln Arg Lys Ala Gln Glu Gly Lys
145 150
<210> 106
<211> 138
<212> БЕЛОК
<213> Ricinus communis
<400> 106
Met Ala Glu His Gln Gln Ser Pro Val Val Ser His Arg Pro Arg Val
1 5 10 15
Asn Gln Leu Val Lys Ala Gly Thr Ala Ala Thr Ala Gly Ser Ser Leu
20 25 30
Leu Phe Leu Ser Gly Leu Thr Leu Thr Gly Thr Val Ile Ala Leu Ala
35 40 45
Leu Ala Thr Pro Leu Met Val Leu Phe Ser Pro Val Leu Leu Pro Ala
50 55 60
Val Ile Ile Ile Ser Leu Ile Gly Ala Gly Phe Leu Thr Ser Gly Gly
65 70 75 80
Phe Gly Phe Gly Ala Ile Leu Val Leu Ser Trp Ile Tyr Arg Tyr Val
85 90 95
Thr Gly Lys Gln Pro Pro Gly Ala Glu Ser Leu Asp Gln Ala Arg Leu
100 105 110
Lys Leu Ala Gly Lys Ala Arg Glu Met Lys Asp Arg Ala Glu Gln Phe
115 120 125
Gly Gln His Val Thr Gly Gln Gln Thr Ser
130 135
<210> 107
<211> 226
<212> БЕЛОК
<213> Glycine max
<400> 107
Met Thr Thr Gln Val Pro Pro His Ser Val Gln Val His Thr Thr Thr
1 5 10 15
Thr His Arg Tyr Glu Ala Gly Val Val Pro Pro Gly Ala Arg Phe Glu
20 25 30
Thr Ser Tyr Glu Ala Gly Val Lys Ala Ala Ser Ile Tyr His Ser Glu
35 40 45
Arg Gly Pro Thr Thr Ser Gln Val Leu Ala Val Leu Ala Gly Leu Pro
50 55 60
Val Gly Gly Ile Leu Leu Leu Leu Ala Gly Leu Thr Leu Ala Gly Thr
65 70 75 80
Leu Thr Gly Leu Ala Val Ala Thr Pro Leu Phe Val Leu Phe Ser Pro
85 90 95
Val Leu Val Pro Ala Thr Val Ala Ile Gly Leu Ala Val Ala Gly Phe
100 105 110
Leu Thr Ser Gly Ala Phe Gly Leu Thr Ala Leu Ser Ser Phe Ser Trp
115 120 125
Ile Leu Asn Tyr Ile Arg Glu Thr Gln Pro Ala Ser Glu Asn Leu Ala
130 135 140
Ala Ala Ala Lys His His Leu Ala Glu Ala Ala Glu Tyr Val Gly Gln
145 150 155 160
Lys Thr Lys Glu Val Gly Gln Lys Thr Lys Glu Val Gly Gln Asp Ile
165 170 175
Gln Ser Lys Ala Gln Asp Thr Arg Glu Ala Ala Ala Arg Asp Ala Arg
180 185 190
Glu Ala Ala Ala Arg Asp Ala Arg Glu Ala Ala Ala Arg Asp Ala Lys
195 200 205
Val Glu Ala Arg Asp Val Lys Arg Thr Thr Val Thr Ala Thr Thr Ala
210 215 220
Thr Ala
225
<210> 108
<211> 223
<212> БЕЛОК
<213> Glycine max
<400> 108
Met Thr Thr Val Pro Pro His Ser Val Gln Val His Thr Thr Thr His
1 5 10 15
Arg Tyr Glu Ala Gly Val Val Pro Pro Ala Arg Phe Glu Ala Pro Arg
20 25 30
Tyr Glu Ala Gly Ile Lys Ala Pro Ser Ser Ile Tyr His Ser Glu Arg
35 40 45
Gly Pro Thr Thr Ser Gln Val Leu Ala Val Val Ala Gly Leu Pro Val
50 55 60
Gly Gly Ile Leu Leu Leu Leu Ala Gly Leu Thr Leu Ala Gly Thr Leu
65 70 75 80
Thr Gly Leu Val Val Ala Thr Pro Leu Phe Ile Ile Phe Ser Pro Val
85 90 95
Leu Ile Pro Ala Thr Val Ala Ile Gly Leu Ala Val Ala Gly Phe Leu
100 105 110
Thr Ser Gly Val Phe Gly Leu Thr Ala Leu Ser Ser Phe Ser Trp Ile
115 120 125
Leu Asn Tyr Ile Arg Glu Thr Gln Pro Ala Ser Glu Asn Leu Ala Ala
130 135 140
Ala Ala Lys His His Leu Ala Glu Ala Ala Glu Tyr Val Gly Gln Lys
145 150 155 160
Thr Lys Glu Val Gly Gln Lys Thr Lys Glu Val Gly Gln Asp Ile Gln
165 170 175
Ser Lys Ala Gln Asp Thr Arg Glu Ala Ala Ala Arg Asp Ala Arg Asp
180 185 190
Ala Arg Glu Ala Ala Ala Arg Asp Ala Arg Asp Ala Lys Val Glu Ala
195 200 205
Arg Asp Val Lys Arg Thr Thr Val Thr Ala Thr Thr Ala Thr Ala
210 215 220
<210> 109
<211> 155
<212> БЕЛОК
<213> Linum usitatissimum
<400> 109
Met Asp Gln Thr His Gln Thr Tyr Ala Gly Thr Thr Gln Asn Pro Ser
1 5 10 15
Tyr Gly Gly Gly Gly Thr Met Tyr Gln Gln Gln Gln Pro Arg Ser Tyr
20 25 30
Gln Ala Val Lys Ala Ala Thr Ala Ala Thr Ala Gly Gly Ser Leu Ile
35 40 45
Val Leu Ser Gly Leu Ile Leu Thr Ala Thr Val Ile Ser Leu Ile Ile
50 55 60
Ala Thr Pro Leu Leu Val Ile Phe Ser Pro Val Leu Val Pro Ala Leu
65 70 75 80
Ile Thr Val Gly Leu Leu Ile Thr Gly Phe Leu Ala Ser Gly Gly Phe
85 90 95
Gly Val Ala Ala Val Thr Val Leu Ser Trp Ile Tyr Arg Tyr Val Thr
100 105 110
Gly Gly His Pro Ala Gly Gly Asp Ser Leu Asp Gln Ala Arg Ser Lys
115 120 125
Leu Ala Gly Lys Ala Arg Glu Val Lys Asp Arg Ala Ser Glu Phe Ala
130 135 140
Gln Gln His Val Thr Gly Gly Gln Gln Thr Ser
145 150 155
<210> 110
<211> 180
<212> БЕЛОК
<213> Linum usitatissimum
<400> 110
Met Ala Asp Arg Thr Thr Gln Pro His Gln Val Gln Val His Thr Gln
1 5 10 15
His His Tyr Pro Thr Gly Gly Ala Phe Gly Arg Tyr Glu Gly Val Leu
20 25 30
Lys Gly Gly Pro Tyr His Gln Gln Gly Thr Gly Ser Gly Pro Ser Ala
35 40 45
Ser Lys Val Leu Ala Val Met Thr Ala Leu Pro Ile Gly Gly Thr Leu
50 55 60
Leu Ala Leu Ala Gly Ile Thr Leu Ala Gly Thr Met Ile Gly Leu Ala
65 70 75 80
Ile Thr Thr Pro Ile Phe Val Ile Cys Ser Pro Val Leu Val Pro Ala
85 90 95
Ala Leu Leu Ile Gly Phe Ala Val Ser Ala Phe Leu Ala Ser Gly Met
100 105 110
Ala Gly Leu Thr Gly Leu Thr Ser Leu Ser Trp Phe Ala Arg Tyr Leu
115 120 125
Gln Gln Ala Gly Gln Gly Val Gly Val Gly Val Pro Asp Ser Phe Asp
130 135 140
Gln Ala Lys Arg Arg Met Gln Asp Ala Ala Gly Tyr Met Gly Gln Lys
145 150 155 160
Thr Lys Glu Val Gly Gln Glu Ile Gln Arg Lys Ser Gln Asp Val Lys
165 170 175
Ala Ser Asp Lys
180
<210> 111
<211> 181
<212> БЕЛОК
<213> Helianthus annuus
<400> 111
Thr Thr Thr Thr Tyr Asp Arg His Phe Thr Thr Thr Gln Pro His Tyr
1 5 10 15
Arg Gln Asp Asp Arg Ser Arg Tyr Asp Gln Gln Thr His Ser Gln Ser
20 25 30
Thr Ser Arg Thr Leu Ala Ile Ile Ala Leu Leu Pro Val Gly Gly Ile
35 40 45
Leu Leu Gly Leu Ala Ala Leu Thr Phe Ile Gly Thr Leu Ile Gly Leu
50 55 60
Ala Leu Ala Thr Pro Leu Phe Val Ile Phe Ser Pro Ile Ile Val Pro
65 70 75 80
Ala Val Leu Thr Ile Gly Leu Ala Val Thr Gly Phe Leu Ala Ser Gly
85 90 95
Thr Phe Gly Leu Thr Gly Leu Ser Ser Leu Ser Tyr Leu Phe Asn Met
100 105 110
Val Arg Gln Thr Ala Gly Ser Val Pro Glu Ser Leu Asp Tyr Val Lys
115 120 125
Gly Thr Leu Gln Asp Ala Gly Glu Tyr Ala Gly Gln Lys Thr Lys Asp
130 135 140
Phe Gly Gln Lys Ile Gln Ser Thr Ala His Glu Met Gly Asp Gln Gly
145 150 155 160
Gln Val Gly Val His Ala Gln Val Gly Gly Gly Lys Glu Gly Arg Lys
165 170 175
Ser Gly Asp Arg Thr
180
<210> 112
<211> 156
<212> БЕЛОК
<213> Zea mays
<400> 112
Met Ala Asp His His Arg Gly Ala Thr Gly Gly Gly Gly Gly Tyr Gly
1 5 10 15
Asp Leu Gln Arg Gly Gly Gly Met His Gly Glu Ala Gln Gln Gln Gln
20 25 30
Lys Gln Gly Ala Met Met Thr Ala Leu Lys Ala Ala Thr Ala Ala Thr
35 40 45
Phe Gly Gly Ser Met Leu Val Leu Ser Gly Leu Ile Leu Ala Gly Thr
50 55 60
Val Ile Ala Leu Thr Val Ala Thr Pro Val Leu Val Ile Phe Ser Pro
65 70 75 80
Val Leu Val Pro Ala Ala Ile Ala Leu Ala Leu Met Ala Ala Gly Phe
85 90 95
Val Thr Ser Gly Gly Leu Gly Val Ala Ala Leu Ser Val Phe Ser Trp
100 105 110
Met Tyr Lys Tyr Leu Thr Gly Lys His Pro Pro Ala Ala Asp Gln Leu
115 120 125
Asp His Ala Lys Ala Arg Leu Ala Ser Lys Ala Arg Asp Val Lys Asp
130 135 140
Ala Ala Gln His Arg Ile Asp Gln Ala Gln Gly Ser
145 150 155
<210> 113
<211> 244
<212> БЕЛОК
<213> Brassica napus
<400> 113
Val Ser Lys Pro Asp Asp Cys Arg Arg Ile Val Asp Glu Thr Ile Ser
1 5 10 15
His Phe Gly Arg Leu Asp His Leu Val Asn Asn Ala Gly Ile Met Gln
20 25 30
Ile Ser Met Phe Glu Asn Ile Glu Glu Ile Thr Arg Thr Arg Ala Val
35 40 45
Met Asp Thr Asn Phe Trp Gly Ser Val Tyr Thr Thr Arg Ala Ala Leu
50 55 60
Pro Tyr Leu Arg Gln Ser Asn Gly Lys Ile Val Ala Met Ser Ser Ser
65 70 75 80
Ala Ala Trp Leu Thr Ala Pro Arg Met Ser Phe Tyr Asn Ala Ser Lys
85 90 95
Ala Ala Leu Leu Asn Phe Phe Glu Thr Leu Arg Ile Glu Leu Gly Ser
100 105 110
Asp Val His Ile Thr Ile Val Thr Pro Gly Tyr Ile Glu Ser Glu Leu
115 120 125
Thr Gln Gly Lys Tyr Phe Ser Gly Glu Gly Glu Leu Val Val Asn Gln
130 135 140
Asp Ile Arg Asp Val Gln Ile Gly Ala Phe Pro Val Thr Ser Val Ser
145 150 155 160
Gly Cys Ala Lys Gly Ile Val Lys Gly Val Cys Arg Lys Gln Arg Tyr
165 170 175
Val Thr Glu Pro Ser Trp Phe Lys Val Thr Tyr Leu Trp Lys Val Phe
180 185 190
Cys Pro Glu Leu Ile Glu Trp Gly Cys Arg Leu Leu Phe Leu Ser Gly
195 200 205
His Gly Thr Ser Glu Lys Asn Ala Leu Asn Lys Lys Ile Leu Asp Ile
210 215 220
Pro Gly Val Arg Ser Ala Leu Tyr Pro Glu Ser Ile Arg Thr Pro Glu
225 230 235 240
Ile Lys Ser Glu
<210> 114
<211> 349
<212> БЕЛОК
<213> Brassica napus
<400> 114
Met Glu Leu Ile Asn Asp Phe Leu Asn Leu Thr Ala Pro Phe Phe Thr
1 5 10 15
Phe Phe Gly Leu Cys Phe Phe Leu Pro Pro Phe Tyr Phe Phe Lys Phe
20 25 30
Val Gln Ser Ile Phe Ser Thr Ile Phe Ser Glu Asn Val Tyr Gly Lys
35 40 45
Val Val Leu Ile Thr Gly Ala Ser Ser Gly Ile Gly Glu Gln Leu Ala
50 55 60
Tyr Glu Tyr Ala Ser Lys Gly Ala Cys Leu Ala Leu Thr Ala Arg Arg
65 70 75 80
Lys Asn Arg Leu Glu Glu Val Ala Glu Ile Ala Arg Glu Val Gly Ser
85 90 95
Pro Asn Val Val Thr Val His Ala Asp Val Ser Lys Pro Asp Asp Cys
100 105 110
Arg Arg Ile Val Asp Glu Thr Ile Ser His Phe Gly Arg Leu Asp His
115 120 125
Leu Val Asn Asn Ala Gly Ile Met Gln Ile Ser Met Phe Glu Asn Ile
130 135 140
Glu Glu Ile Thr Arg Thr Arg Ala Val Met Asp Thr Asn Phe Trp Gly
145 150 155 160
Ala Val Tyr Thr Thr Arg Ala Ala Leu Pro Tyr Leu Arg Gln Ser Asn
165 170 175
Gly Lys Ile Val Ala Met Ser Ser Ser Ala Ala Trp Leu Thr Ala Pro
180 185 190
Arg Met Ser Phe Tyr Asn Ala Ser Lys Ala Ala Leu Leu Asn Phe Phe
195 200 205
Glu Thr Leu Arg Ile Glu Leu Gly Ser Asp Val His Ile Thr Ile Val
210 215 220
Thr Pro Gly Tyr Ile Glu Ser Glu Leu Thr Gln Gly Lys Tyr Val Ser
225 230 235 240
Gly Glu Gly Glu Leu Val Val Asn Gln Asp Ile Arg Asp Val Gln Ile
245 250 255
Gly Ala Phe Pro Val Thr Ser Val Ser Gly Arg Ala Lys Gly Ile Val
260 265 270
Lys Gly Val Cys Arg Lys Glu Arg Tyr Val Thr Glu Pro Ser Trp Phe
275 280 285
Lys Val Thr Tyr Leu Trp Lys Val Phe Cys Pro Glu Leu Ile Glu Trp
290 295 300
Gly Cys Arg Leu Met Phe Leu Ser Gly His Gly Thr Pro Glu Glu Asn
305 310 315 320
Ala Leu Asn Lys Lys Ile Leu Asp Ile Pro Gly Val Arg Ser Ala Leu
325 330 335
Tyr Pro Glu Pro Ile Arg Thr Pro Glu Ile Lys Ser Glu
340 345
<210> 115
<211> 456
<212> БЕЛОК
<213> Brassica napus
<400> 115
Met Val Asp Leu Leu Asn Ser Val Met Asn Leu Val Ala Pro Pro Ala
1 5 10 15
Thr Met Val Val Met Ala Phe Ser Trp Pro Leu Leu Cys Phe Ile Thr
20 25 30
Phe Ser Glu Arg Leu Tyr Asn Ser Tyr Phe Val Thr Glu Asp Met Glu
35 40 45
Asp Lys Val Val Val Ile Thr Gly Ala Ser Pro Ala Ile Gly Glu Gln
50 55 60
Ile Ala Tyr Glu Tyr Ala Lys Arg Gly Ala Asn Leu Val Leu Val Ala
65 70 75 80
Arg Arg Glu Gln Arg Leu Arg Val Val Ser Asn Asn Ala Arg Gln Ile
85 90 95
Gly Ala Asn His Val Ile Ile Ile Ala Ala Asp Val Val Lys Glu Asp
100 105 110
Asp Cys Arg Arg Phe Ile Thr Gln Ala Val Asn Tyr Tyr Gly Arg Val
115 120 125
Asp His Leu Val Asn Ser Ala Ser Leu Gly His Thr Phe Tyr Phe Asp
130 135 140
Glu Val Ser Asp Thr Thr Val Phe Pro His Leu Leu Asp Ile Asn Phe
145 150 155 160
Trp Gly Asn Val Tyr Pro Thr Tyr Val Ala Leu Pro His Leu Gln Lys
165 170 175
Thr Asn Gly Arg Ile Val Val Asn Ala Ser Val Glu Asn Trp Leu Pro
180 185 190
Leu Pro Arg Met Ser Leu Tyr Ser Ala Ala Lys Ala Ala Leu Val Asn
195 200 205
Phe Tyr Glu Thr Leu Arg Phe Glu Leu Asn Gly Asp Val Gly Ile Thr
210 215 220
Ile Ala Thr His Gly Trp Ile Gly Ser Glu Met Ser Arg Gly Lys Phe
225 230 235 240
Met Leu Glu Glu Gly Ala Glu Met Gln Trp Lys Glu Glu Arg Glu Val
245 250 255
Pro Ala Asn Gly Gly Pro Leu Glu Glu Phe Ala Lys Met Ile Val Ala
260 265 270
Gly Ala Cys Arg Gly Asp Ala Tyr Val Lys Phe Pro Asn Trp Tyr Asp
275 280 285
Val Phe Leu Leu Tyr Arg Val Phe Thr Pro Asn Val Leu Arg Trp Thr
290 295 300
Phe Lys Leu Leu Leu Ser Ser Glu Gly Ser Arg Gln Ser Ser Leu Val
305 310 315 320
Gly Val Gly Gln Gly Leu Pro Pro Glu Glu Ser Ser Ser Gln Met Lys
325 330 335
Leu Met Leu Glu Gly Gly Ser Pro Arg Val Thr Ala Ser Pro Pro Arg
340 345 350
Tyr Thr Pro Ser Pro Ser Pro Pro His His Thr Ala Ser Pro Pro Arg
355 360 365
Tyr Thr Pro Ser Pro Ser Pro Pro His His Thr Ser Ser Pro Gln Arg
370 375 380
Tyr Thr Pro Ser Pro Ser Pro Pro His Tyr Thr Ser Ser Arg His Arg
385 390 395 400
Tyr Thr Pro Ser Pro Ser Pro Pro His Tyr Thr Glu Ser Pro Pro Leu
405 410 415
Tyr Thr Glu Ser Pro Pro His Tyr Thr Thr Ser Pro Asn Trp Tyr Thr
420 425 430
Glu Ser Pro Pro Arg Tyr Thr Pro Ser Pro Ser Pro Pro Arg Phe Ser
435 440 445
Arg Phe Asn Ile Gln Glu Leu Pro
450 455
<210> 116
<211> 348
<212> БЕЛОК
<213> Sesamum indicum
<400> 116
Met Asp Leu Ile His Thr Phe Leu Asn Leu Ile Ala Pro Pro Phe Thr
1 5 10 15
Phe Phe Phe Leu Leu Phe Phe Leu Pro Pro Phe Gln Ile Phe Lys Phe
20 25 30
Phe Leu Ser Ile Leu Gly Thr Leu Phe Ser Glu Asp Val Ala Gly Lys
35 40 45
Val Val Val Ile Thr Gly Ala Ser Ser Gly Ile Gly Glu Ser Leu Ala
50 55 60
Tyr Glu Tyr Ala Lys Arg Gly Ala Cys Leu Val Leu Ala Ala Arg Arg
65 70 75 80
Glu Arg Ser Leu Gln Glu Val Ala Glu Arg Ala Arg Asp Leu Gly Ser
85 90 95
Pro Asp Val Val Val Val Arg Ala Asp Val Ser Lys Ala Glu Asp Cys
100 105 110
Arg Lys Val Val Asp Gln Thr Met Asn Arg Phe Gly Arg Leu Asp His
115 120 125
Leu Val Asn Asn Ala Gly Ile Met Ser Val Ser Met Leu Glu Glu Val
130 135 140
Glu Asp Ile Thr Gly Tyr Arg Glu Thr Met Asp Ile Asn Phe Trp Gly
145 150 155 160
Tyr Val Tyr Met Thr Arg Phe Ala Ala Pro Tyr Leu Arg Asn Ser Arg
165 170 175
Gly Arg Ile Val Val Leu Ser Ser Ser Ser Ser Trp Met Pro Thr Pro
180 185 190
Arg Met Ser Phe Tyr Asn Ala Ser Lys Ala Ala Ile Ser Gln Phe Phe
195 200 205
Glu Thr Leu Arg Val Glu Phe Gly Pro Asp Ile Gly Ile Thr Leu Val
210 215 220
Thr Pro Gly Phe Ile Glu Ser Glu Leu Thr Gln Gly Lys Phe Tyr Asn
225 230 235 240
Ala Gly Glu Arg Val Ile Asp Gln Asp Met Arg Asp Val Gln Val Ser
245 250 255
Thr Thr Pro Ile Leu Arg Val Glu Ser Ala Ala Arg Ser Ile Val Arg
260 265 270
Ser Ala Ile Arg Gly Glu Arg Tyr Val Thr Glu Pro Ala Trp Phe Arg
275 280 285
Val Thr Tyr Trp Trp Lys Leu Phe Cys Pro Glu Val Met Glu Trp Val
290 295 300
Phe Arg Leu Met Tyr Leu Ala Ser Pro Gly Glu Pro Glu Lys Glu Thr
305 310 315 320
Phe Gly Lys Lys Val Leu Asp Tyr Thr Gly Val Lys Ser Leu Leu Tyr
325 330 335
Pro Glu Thr Val Gln Val Pro Glu Pro Lys Asn Asp
340 345
<210> 117
<211> 350
<212> БЕЛОК
<213> Zea mays
<400> 117
Met Leu Gly Met Ser Arg Thr Gly Leu Ala Gly Ala Ala Leu Arg Val
1 5 10 15
Ala Leu Thr Ala Leu Leu Pro Leu Val Leu Pro Ala Tyr Tyr Val Tyr
20 25 30
Lys Leu Thr Thr Tyr Leu Leu Gly Ala Val Phe Pro Glu Asp Val Ala
35 40 45
Gly Lys Val Val Leu Ile Thr Gly Ala Ser Ser Gly Ile Gly Glu His
50 55 60
Leu Ala Tyr Glu Tyr Ala Lys Arg Gly Ala Tyr Leu Ala Leu Val Ala
65 70 75 80
Arg Arg Glu Ala Ser Leu Arg Glu Val Gly Asp Val Ala Leu Gly Leu
85 90 95
Gly Ser Pro Gly Val Leu Val Leu Pro Ala Asp Val Ser Lys Pro Arg
100 105 110
Asp Cys Glu Gly Phe Ile Asp Asp Thr Ile Ser Tyr Phe Gly Arg Leu
115 120 125
Asp His Leu Val Asn Asn Ala Ser Ile Trp Gln Val Cys Lys Phe Glu
130 135 140
Glu Ile Gln Asp Val Arg His Leu Arg Ala Leu Met Asp Ile Asn Phe
145 150 155 160
Trp Gly His Val Tyr Pro Thr Arg Leu Ala Ile Pro His Leu Arg Arg
165 170 175
Ser Arg Gly Arg Ile Val Gly Val Thr Ser Asn Ser Ser Tyr Ile Phe
180 185 190
Ile Gly Arg Asn Thr Phe Tyr Asn Ala Ser Lys Ala Ala Ala Leu Ser
195 200 205
Phe Tyr Asp Thr Leu Arg Met Glu Leu Gly Ser Asp Ile Arg Ile Thr
210 215 220
Glu Val Val Pro Gly Val Val Glu Ser Glu Ile Thr Lys Gly Lys Met
225 230 235 240
Leu Thr Lys Gly Gly Glu Met Lys Val Asp Gln Asp Glu Arg Asp Ala
245 250 255
Ile Leu Gly Pro Thr Pro Ala Glu Pro Val Gly Asp Phe Ala Arg Thr
260 265 270
Val Val Arg Asp Val Cys Arg Gly Ala Arg Tyr Val Phe Glu Pro Arg
275 280 285
Trp Tyr Met Gly Val Tyr Leu Leu Arg Ala Cys Leu Pro Glu Val Leu
290 295 300
Ala Trp Asn Ser Arg Leu Leu Thr Val Asp Thr Val Gly Ala Ser Thr
305 310 315 320
Thr Asp Thr Leu Gly Lys Trp Leu Val Glu Leu Pro Gly Val Arg Arg
325 330 335
Val Val Gln Pro Pro Ser Leu Arg Ser Pro Glu Ile Lys Asp
340 345 350
<210> 118
<211> 245
<212> БЕЛОК
<213> Brassica napus
<400> 118
Met Gly Thr Ala Thr Glu Ile Met Glu Arg Asp Ala Met Ala Thr Val
1 5 10 15
Ala Pro Tyr Ala Pro Val Thr Phe His Arg Arg Ala Arg Val Asp Leu
20 25 30
Asp Asp Arg Leu Pro Lys Pro Tyr Met Pro Arg Ala Leu Gln Ala Pro
35 40 45
Asp Arg Glu His Pro Tyr Gly Thr Pro Gly His Lys Asn Tyr Gly Leu
50 55 60
Ser Val Leu Gln Gln His Val Ala Phe Phe Asp Ile Asp Asp Asn Gly
65 70 75 80
Ile Ile Tyr Pro Trp Glu Thr Tyr Ser Gly Leu Arg Met Ile Gly Phe
85 90 95
Asn Ile Ile Gly Ser Leu Ile Ile Ala Ala Val Ile Asn Leu Ala Leu
100 105 110
Ser Tyr Ala Thr Leu Pro Gly Trp Leu Pro Ser Pro Phe Phe Pro Ile
115 120 125
Tyr Ile His Asn Ile His Lys Ser Lys His Gly Ser Asp Ser Arg Thr
130 135 140
Tyr Asp Asn Glu Gly Arg Phe Met Pro Val Asn Leu Glu Leu Ile Phe
145 150 155 160
Ser Lys Tyr Ala Lys Thr Leu Pro Asp Lys Leu Ser Leu Gly Glu Leu
165 170 175
Trp Asp Met Thr Glu Gly Gln Arg Asp Ala Trp Asp Ile Phe Gly Trp
180 185 190
Phe Ala Ser Lys Ile Glu Trp Gly Leu Leu Tyr Leu Leu Ala Arg Asp
195 200 205
Glu Glu Gly Phe Leu Ser Lys Glu Ala Ile Arg Arg Cys Phe Asp Gly
210 215 220
Ser Leu Phe Glu Tyr Cys Ala Lys Ile Tyr Val Gly Ile Asn Glu Asp
225 230 235 240
Lys Thr Ala Tyr Tyr
245
<210> 119
<211> 244
<212> БЕЛОК
<213> Brassica napus
<400> 119
Met Val Arg Glu Ser Met Gly Glu Glu Ser Glu Ala Phe Ala Thr Thr
1 5 10 15
Ala Pro Leu Ala Pro Val Thr Gly Glu Arg Lys Val Arg Asn Asp Leu
20 25 30
Glu Glu Thr Leu Pro Lys Pro Tyr Leu Ala Arg Ala Leu Val Ala Pro
35 40 45
Asp Thr Glu His Pro Asn Gly Ser Glu Gly His Asp Ser Lys Gly Met
50 55 60
Ser Val Thr Gln Gln His Val Ala Phe Phe Asp Gln Asn Gly Asp Gly
65 70 75 80
Ile Val Tyr Pro Trp Glu Thr Tyr Ala Gly Phe Arg Asp Leu Gly Phe
85 90 95
Asn Pro Ile Ser Ser Val Phe Trp Ala Ile Phe Ile Asn Phe Ala Phe
100 105 110
Ser Tyr Val Thr Leu Pro Ser Trp Leu Pro Ser Pro Leu Leu Pro Val
115 120 125
Tyr Ile Asp Asn Ile His Lys Ala Lys His Gly Ser Asp Ser Ser Thr
130 135 140
Tyr Asp Thr Glu Gly Arg Tyr Val Pro Val Asn Leu Glu Asn Ile Phe
145 150 155 160
Ser Lys Tyr Ala Leu Thr Ala Pro Asn Lys Ile Thr Leu Lys Glu Leu
165 170 175
Trp Asn Leu Thr Glu Gly Asn Arg Met Ala Ile Asp Pro Phe Gly Trp
180 185 190
Leu Ala Asn Lys Val Glu Trp Leu Leu Val Tyr Leu Leu Ala Lys Asp
195 200 205
Glu Glu Gly Phe Val Ser Lys Glu Ala Val Arg Gly Val Phe Asp Ala
210 215 220
Ser Phe Phe Glu Tyr Cys Ala Lys Lys Asn Lys Glu Lys Ala Asp Ser
225 230 235 240
Arg Lys Gln Asp
<210> 120
<211> 245
<212> БЕЛОК
<213> Sesamum indicum
<400> 120
Met Ala Thr His Val Leu Ala Ala Ala Ala Glu Arg Asn Ala Ala Leu
1 5 10 15
Ala Pro Asp Ala Pro Leu Ala Pro Val Thr Met Glu Arg Pro Val Arg
20 25 30
Thr Asp Leu Glu Thr Ser Ile Pro Lys Pro Tyr Met Ala Arg Gly Leu
35 40 45
Val Ala Pro Asp Met Asp His Pro Asn Gly Thr Pro Gly His Val His
50 55 60
Asp Asn Leu Ser Val Leu Gln Gln His Cys Ala Phe Phe Asp Gln Asp
65 70 75 80
Asp Asn Gly Ile Ile Tyr Pro Trp Glu Thr Tyr Ser Gly Leu Arg Gln
85 90 95
Ile Gly Phe Asn Val Ile Ala Ser Leu Ile Met Ala Ile Val Ile Asn
100 105 110
Val Ala Leu Ser Tyr Pro Thr Leu Pro Gly Trp Ile Pro Ser Pro Phe
115 120 125
Phe Pro Ile Tyr Leu Tyr Asn Ile His Lys Ala Lys His Gly Ser Asp
130 135 140
Ser Gly Thr Tyr Asp Thr Glu Gly Arg Tyr Leu Pro Met Asn Phe Glu
145 150 155 160
Asn Leu Phe Ser Lys His Ala Arg Thr Met Pro Asp Arg Leu Thr Leu
165 170 175
Gly Glu Leu Trp Ser Met Thr Glu Ala Asn Arg Glu Ala Phe Asp Ile
180 185 190
Phe Gly Trp Ile Ala Ser Lys Met Glu Trp Thr Leu Leu Tyr Ile Leu
195 200 205
Ala Arg Asp Gln Asp Gly Phe Leu Ser Lys Glu Ala Ile Arg Arg Cys
210 215 220
Tyr Asp Gly Ser Leu Phe Glu Tyr Cys Ala Lys Met Gln Arg Gly Ala
225 230 235 240
Glu Asp Lys Met Lys
245
<210> 121
<211> 243
<212> БЕЛОК
<213> Zea mays
<400> 121
Met Ser Ser Tyr Ser Pro Pro Pro Pro Pro Pro Arg Asp Gln Ser Met
1 5 10 15
Asp Thr Glu Ala Pro Asn Ala Pro Ile Thr Arg Glu Arg Arg Leu Asn
20 25 30
Pro Asp Leu Gln Glu Gln Leu Pro Lys Pro Tyr Leu Ala Arg Ala Leu
35 40 45
Glu Ala Val Asp Pro Ser His Pro Gln Gly Thr Lys Gly Arg Asp Pro
50 55 60
Arg Gly Met Ser Val Leu Gln Gln His Ala Ala Phe Phe Asp Arg Asn
65 70 75 80
Gly Asp Gly Val Ile Tyr Pro Trp Glu Thr Phe Gln Gly Leu Arg Ala
85 90 95
Ile Gly Cys Gly Leu Thr Val Ser Phe Ala Phe Ser Ile Leu Ile Asn
100 105 110
Leu Phe Leu Ser Tyr Pro Thr Gln Pro Gly Trp Leu Pro Ser Pro Leu
115 120 125
Leu Ser Ile Arg Ile Asp Asn Ile His Lys Gly Lys His Gly Ser Asp
130 135 140
Ser Glu Thr Tyr Asp Thr Glu Gly Arg Phe Asp Pro Ser Lys Phe Asp
145 150 155 160
Ala Ile Phe Ser Lys Tyr Gly Arg Thr His Pro Asn Ala Ile Thr Arg
165 170 175
Asp Glu Leu Ser Ser Met Leu Gln Gly Asn Arg Asn Thr Tyr Asp Phe
180 185 190
Leu Gly Trp Leu Ala Ala Ala Gly Glu Trp Leu Leu Leu Tyr Ser Leu
195 200 205
Ala Lys Asp Lys Asp Gly Leu Leu Gln Arg Glu Thr Val Arg Gly Leu
210 215 220
Phe Asp Gly Ser Leu Phe Glu Arg Leu Glu Asp Asp Asn Asn Lys Lys
225 230 235 240
Lys Ser Ser
<210> 122
<211> 11142
<212> ДНК
<213> Искусственная последовательность
<220>
<223> последовательность T-ДНК
<400> 122
tcctgtggtt ggcatgcaca tacaaatgga cgaacggata aaccttttca cgccctttta
60
aatatccgat tattctaata aacgctcttt tctcttaggt ttacccgcca atatatcctg
120
tcaaacactg atagtttaaa ctgaaggcgg gaaacgacaa tctgctagtg gatctcccag
180
tcacgacgtt gtaaaacggg cgccctagaa tctaattatt ctattcagac taaattagta
240
taagtatttt tttaatcaat aaataataat taataattta ttagtaggag tgattgaatt
300
tataatatat tttttttaat catttaaaga atcttatatc tttaaattga caagagtttt
360
aaatggggag agtgttatca tatcacaagt aggattaatg tgttatagtt tcacatgcat
420
tacgataagt tgtgaaagat aacattatta tatataacaa tgacaatcac tagcgatcga
480
gtagtgagag tcgtcttatt acactttctt ccttcgatct gtcacatggc ggcggcccga
540
attctcacac aaggtagttg caagacactg aagtggtggt agtggtagta gaagaagcag
600
aatcggtaga aaggcaagac aatggagaag atgaagatgg tggagattct cttcccacaa
660
cgcagcaatc aaggttttca aggttaaggc actcgtgctt tccatcatcg aacatgaagt
720
cgatgttatc ctcgaaagca agctcgttga agagttctgg gtactcaatt gggttctcgt
780
tagcaaggtt ttgatcggta aggaatgggg agaatccagt atccatcatg cagaagttcc
840
aagcaagttc gttgttatct ccgcacctat ccatttccat gatggtggaa gaatcaatgc
900
agcagttaac aacggcagct tcctcagaat atcccacaat ttcagcctct tgttgctcag
960
ccttctcttc ctctttttct tcttcctctt gaggtggttc ctcaacgtat tgttgcttaa
1020
cctcttccct aggttcctct ttagcttctc tagtctcaac ctcttgctta gcctcaacaa
1080
gaataccctc ttgatggtta gcctggttaa ctgggaatgg gaaaacgccc ttcttcttaa
1140
gcctgtcgat gtagttggag atatcgaagt tggtaacagc gttagcacct ctgtactcaa
1200
tagcagccat atcataagca gctgcagcct cttcttgagt gttgtaagtt ccgaggtaga
1260
ggtacttgtt tccgaaaact cttccaatcc tagcttccca tcttccgtta tgatgatgcc
1320
tagcaactcc cctatactta gaaactcccc tagagaatcc agatgactgc cttctaaggg
1380
aagcaagata ctcttctttg gtcaccctct gcatctcttc aagttctttg gtgtaagtct
1440
cagctgggaa gttaagaatg gtatctgggc cccaatactt aagagcagca agatcatagg
1500
tatgagcagc agcctcttca gaatcataag ctccaaggta aacctgcttg cccttcttgt
1560
tttggatgga gttccaagag gacttatccc aaaggtgagc ttcgaatctt ccagtccatc
1620
tatgcctagt aacacctctg tagatagatg accttctggt agaagctgga gaagttgggt
1680
tatgagactt atcgccagat ggagatgact tcttagccct cttagctctc tttggtcttg
1740
gagcttcaga ttgaattggg ctagaggtag tagtagaaga ggacactgaa gaagatggag
1800
aactagagca ggtagaggta gtgagcctct tcttcatgaa ttctgttctt ctttactctt
1860
tgtgtgactg aggtttggtc tagtgctttg gtcatctata tataatgata acaacaatga
1920
gaacaagctt tggagtgatc ggagggtcta ggatacatga gattcaagtg gactaggatc
1980
tacaccgttg gattttgagt gtggatatgt gtgaggttaa ttttacttgg taacggccac
2040
aaaggcctaa ggagaggtgt tgagaccctt atcggcttga accgctggaa taatgccacg
2100
tggaagataa ttccatgaat cttatcgtta tctatgagtg aaattgtgtg atggtggagt
2160
ggtgcttgct cattttactt gcctggtgga cttggccctt tccttatggg gaatttatat
2220
tttacttact atagagcttt catacctttt ttttaccttg gatttagtta atatataatg
2280
gtatgattca tgaataaaaa tgggaaattt ttgaatttgt actgctaaat gcataagatt
2340
aggtgaaact gtggaatata tatttttttc atttaaaagc aaaatttgcc ttttactaga
2400
attataaata tagaaaaata tataacattc aaataaaaat gaaaataaga actttcaaaa
2460
aacagaacta tgtttaatgt gtaaagatta gtcgcacatc aagtcatctg ttacaatatg
2520
ttacaacaag tcataagccc aacaaagtta gcacgtctaa ataaactaaa gagtccacga
2580
aaatattaca aatcataagc ccaacaaagt tattgatcaa aaaaaaaaaa cgcccaacaa
2640
agctaaacaa agtccaaaaa aaacttctca agtctccatc ttcctttatg aacattgaaa
2700
actatacaca aaacaagtca gataaatctc tttctgggcc tgtcttccca acctcctaca
2760
tcacttccct atcggattga atgttttact tgtacctttt ccgttgcaat gatattgata
2820
gtatgtttgt gaaaactaat agggttaaca atcgaagtca tggaatatgg atttggtcca
2880
agattttccg agagctttct agtagaaagc ccatcaccag aaatttacta gtaaaataaa
2940
tcaccaatta ggtttcttat tatgtgccaa attcaatata attatagagg atatttcaaa
3000
tgaaaacgta tgaatgttat tagtaaatgg tcaggtaaga cattaaaaaa atcctacgtc
3060
agatattcaa ctttaaaaat tcgatcagtg tggaattgta caaaaatttg ggatctacta
3120
tatatatata atgctttaca acacttggat ttttttttgg aggctggaat ttttaatcta
3180
catatttgtt ttggccatgc accaactcat tgtttagtgt aatactttga ttttgtcaaa
3240
tatatgtgtt cgtgtatatt tgtataagaa tttctttgac catatacaca cacacatata
3300
tatatatata tatatattat atatcatgca cttttaattg aaaaaataat atatatatat
3360
atagtgcatt ttttctaaca accatatatg ttgcgattga tctgcaaaaa tactgctaga
3420
gtaatgaaaa atataatcta ttgctgaaat tatctcagat gttaagattt tcttaaagta
3480
aattctttca aattttagct aaaagtcttg taataactaa agaataatac acaatctcga
3540
ccacggaaaa aaaacacata ataaatttgg ggcccctaga atctaattat tctattcaga
3600
ctaaattagt ataagtattt ttttaatcaa taaataataa ttaataattt attagtagga
3660
gtgattgaat ttataatata ttttttttaa tcatttaaag aatcttatat ctttaaattg
3720
acaagagttt taaatgggga gagtgttatc atatcacaag taggattaat gtgttatagt
3780
ttcacatgca ttacgataag ttgtgaaaga taacattatt atatataaca atgacaatca
3840
ctagcgatcg agtagtgaga gtcgtcttat tacactttct tccttcgatc tgtcacatgg
3900
cggcggcccg cggccgcttc attactcgag ccaggaggat ggatcgatgc tggtctgaga
3960
ccctgctacc ggttgctgac tgaactgctc ggcacggtcc ttcatttcac gggccttgct
4020
cgccaacttt gtcttggccg actccaactg atccgctccg ggtggatgtt tccccgtcag
4080
gtaacggtag atccaggaca gcacagacag agcggcaaca ccaaatcccc cgcttgccag
4140
aaaacccgct cccaacagga agatggtgat gactgcagat cagaaaaact cagattaatc
4200
gacaaattcg atcgcacaaa ctagaaacta acaccagatc tagatagaaa tcacaaatcg
4260
aagagtaatt attcgacaaa actcaaatta tttgaacaaa tcggatgata tctatgaaac
4320
cctaatcgag aattaagatg atatctaacg atcaaaccca gaaaatcgtc ttcgatctaa
4380
gattaacaga atctaaacca aagaacatat acgaaattgg gatcgaacga aaacaaaatc
4440
gaagattttg agagaataag gaacacagaa atttacctgc agggaccagt acaggcgaga
4500
agatcaccag gagaggtgtg gcgattgtca gcgcaatgac cgttccagcc agggtcaacc
4560
cggataacac caacaggcta cctccggcag taaccgcggt cgctgccttt acaacacgct
4620
gagcacgcgg ttgcagttgc aagtgggggg cacgtgtttg ttgctgctgc ccgtagtgct
4680
ctgccatggt tttttttaac ggagcaagcg gccgctgttc ttctttactc tttgtgtgac
4740
tgaggtttgg tctagtgctt tggtcatcta tatataatga taacaacaat gagaacaagc
4800
tttggagtga tcggagggtc taggatacat gagattcaag tggactagga tctacaccgt
4860
tggattttga gtgtggatat gtgtgaggtt aattttactt ggtaacggcc acaaaggcct
4920
aaggagaggt gttgagaccc ttatcggctt gaaccgctgg aataatgcca cgtggaagat
4980
aattccatga atcttatcgt tatctatgag tgaaattgtg tgatggtgga gtggtgcttg
5040
ctcattttac ttgcctggtg gacttggccc tttccttatg gggaatttat attttactta
5100
ctatagagct ttcatacctt ttttttacct tggatttagt taatatataa tggtatgatt
5160
catgaataaa aatgggaaat ttttgaattt gtactgctaa atgcataaga ttaggtgaaa
5220
ctgtggaata tatatttttt tcatttaaaa gcaaaatttg ccttttacta gaattataaa
5280
tatagaaaaa tatataacat tcaaataaaa atgaaaataa gaactttcaa aaaacagaac
5340
tatgtttaat gtgtaaagat tagtcgcaca tcaagtcatc tgttacaata tgttacaaca
5400
agtcataagc ccaacaaagt tagcacgtct aaataaacta aagagtccac gaaaatatta
5460
caaatcataa gcccaacaaa gttattgatc aaaaaaaaaa aacgcccaac aaagctaaac
5520
aaagtccaaa aaaaacttct caagtctcca tcttccttta tgaacattga aaactataca
5580
caaaacaagt cagataaatc tctttctggg cctgtcttcc caacctccta catcacttcc
5640
ctatcggatt gaatgtttta cttgtacctt ttccgttgca atgatattga tagtatgttt
5700
gtgaaaacta atagggttaa caatcgaagt catggaatat ggatttggtc caagattttc
5760
cgagagcttt ctagtagaaa gcccatcacc agaaatttac tagtaaaata aatcaccaat
5820
taggtttctt attatgtgcc aaattcaata taattataga ggatatttca aatgaaaacg
5880
tatgaatgtt attagtaaat ggtcaggtaa gacattaaaa aaatcctacg tcagatattc
5940
aactttaaaa attcgatcag tgtggaattg tacaaaaatt tgggatctac tatatatata
6000
taatgcttta caacacttgg attttttttt ggaggctgga atttttaatc tacatatttg
6060
ttttggccat gcaccaactc attgtttagt gtaatacttt gattttgtca aatatatgtg
6120
ttcgtgtata tttgtataag aatttctttg accatataca cacacacata tatatatata
6180
tatatatatt atatatcatg cacttttaat tgaaaaaata atatatatat atatagtgca
6240
ttttttctaa caaccatata tgttgcgatt gatctgcaaa aatactgcta gagtaatgaa
6300
aaatataatc tattgctgaa attatctcag atgttaagat tttcttaaag taaattcttt
6360
caaattttag ctaaaagtct tgtaataact aaagaataat acacaatctc gaccacggaa
6420
aaaaaacaca taataaattt gggcgcgccg cgtattggct agagcagctt gccaacatgg
6480
tggagcacga cactctcgtc tactccaaga atatcaaaga tacagtctca gaagaccaaa
6540
gggctattga gacttttcaa caaagggtaa tatcgggaaa cctcctcgga ttccattgcc
6600
cagctatctg tcacttcatc aaaaggacag tagaaaagga aggtggcacc tacaaatgcc
6660
atcattgcga taaaggaaag gctatcgttc aagatgcctc tgccgacagt ggtcccaaag
6720
atggaccccc acccacgagg agcatcgtgg aaaaagaaga cgttccaacc acgtcttcaa
6780
agcaagtgga ttgatgtgat aacatggtgg agcacgacac tctcgtctac tccaagaata
6840
tcaaagatac agtctcagaa gaccaaaggg ctattgagac ttttcaacaa agggtaatat
6900
cgggaaacct cctcggattc cattgcccag ctatctgtca cttcatcaaa aggacagtag
6960
aaaaggaagg tggcacctac aaatgccatc attgcgataa aggaaaggct atcgttcaag
7020
atgcctctgc cgacagtggt cccaaagatg gacccccacc cacgaggagc atcgtggaaa
7080
aagaagacgt tccaaccacg tcttcaaagc aagtggattg atgtgatatc tccactgacg
7140
taagggatga cgcacaatcc cactatcctt cgcaagacct tcctctatat aaggaagttc
7200
atttcatttg gagaggacac gctgaaatca ccagtctctc tctacaaatc tatctctgcg
7260
atcgcatggc gattttggat tctgctggcg ttactacggt gacggagaac ggtggcggag
7320
agttcgtcga tcttgatagg cttcgtcgac ggaaatcgag atcggattct tctaacggac
7380
ttcttctctc tggttccgat aataattctc cttcggatga tgttggagct cccgccgacg
7440
ttagggatcg gattgattcc gttgttaacg atgacgctca gggaacagcc aatttggccg
7500
gagataataa cggtggtggc gataataacg gtggtggaag aggcggcgga gaaggaagag
7560
gaaacgccga tgctacgttt acgtatcgac cgtcggttcc agctcatcgg agggcgagag
7620
agagtccact tagctccgac gcaatcttca aacagagcca tgccggatta ttcaacctct
7680
gtgtagtagt tcttattgct gtaaacagta gactcatcat cgaaaatctt atgaagtatg
7740
gttggttgat cagaacggat ttctggttta gttcaagatc gctgcgagat tggccgcttt
7800
tcatgtgttg tatatccctt tcgatctttc ctttggctgc ctttacggtt gagaaattgg
7860
tacttcagaa atacatatca gaacctgttg tcatctttct tcatattatt atcaccatga
7920
cagaggtttt gtatccagtt tacgtcaccc taaggtgtga ttctgctttt ttatcaggtg
7980
tcactttgat gctcctcact tgcattgtgt ggctaaagtt ggtttcttat gctcatacta
8040
gctatgacat aagatcccta gccaatgcag ctgataaggc caatcctgaa gtctcctact
8100
acgttagctt gaagagcttg gcatatttca tggtcgctcc cacattgtgt tatcagccaa
8160
gttatccacg ttctgcatgt atacggaagg gttgggtggc tcgtcaattt gcaaaactgg
8220
tcatattcac cggattcatg ggatttataa tagaacaata tataaatcct attgtcagga
8280
actcaaagca tcctttgaaa ggcgatcttc tatatgctat tgaaagagtg ttgaagcttt
8340
cagttccaaa tttatatgtg tggctctgca tgttctactg cttcttccac ctttggttaa
8400
acatattggc agagcttctc tgcttcgggg atcgtgaatt ctacaaagat tggtggaatg
8460
caaaaagtgt gggagattac tggagaatgt ggaatatgcc tgttcataaa tggatggttc
8520
gacatatata cttcccgtgc ttgcgcagca agataccaaa gacactcgcc attatcattg
8580
ctttcctagt ctctgcagtc tttcatgagc tatgcatcgc agttccttgt cgtctcttca
8640
agctatgggc ttttcttggg attatgtttc aggtgccttt ggtcttcatc acaaactatc
8700
tacaggaaag gtttggctca acggtgggga acatgatctt ctggttcatc ttctgcattt
8760
tcggacaacc gatgtgtgtg cttctttatt accacgacct gatgaaccga aaaggatcga
8820
tgtcatgagc gatcgcgatc gttcaaacat ttggcaataa agtttcttaa gattgaatcc
8880
tgttgccggt cttgcgatga ttatcatata atttctgttg aattacgtta agcatgtaat
8940
aattaacatg taatgcatga cgttatttat gagatgggtt tttatgatta gagtcccgca
9000
attatacatt taatacgcga tagaaaacaa aatatagcgc gcaaactagg ataaattatc
9060
gcgcgcggtg tcatctatgt tactagatcc ctgcagggcg tattggctag agcagcttgc
9120
caacatggtg gagcacgaca ctctcgtcta ctccaagaat atcaaagata cagtctcaga
9180
agaccaaagg gctattgaga cttttcaaca aagggtaata tcgggaaacc tcctcggatt
9240
ccattgccca gctatctgtc acttcatcaa aaggacagta gaaaaggaag gtggcaccta
9300
caaatgccat cattgcgata aaggaaaggc tatcgttcaa gatgcctctg ccgacagtgg
9360
tcccaaagat ggacccccac ccacgaggag catcgtggaa aaagaagacg ttccaaccac
9420
gtcttcaaag caagtggatt gatgtgataa catggtggag cacgacactc tcgtctactc
9480
caagaatatc aaagatacag tctcagaaga ccaaagggct attgagactt ttcaacaaag
9540
ggtaatatcg ggaaacctcc tcggattcca ttgcccagct atctgtcact tcatcaaaag
9600
gacagtagaa aaggaaggtg gcacctacaa atgccatcat tgcgataaag gaaaggctat
9660
cgttcaagat gcctctgccg acagtggtcc caaagatgga cccccaccca cgaggagcat
9720
cgtggaaaaa gaagacgttc caaccacgtc ttcaaagcaa gtggattgat gtgatatctc
9780
cactgacgta agggatgacg cacaatccca ctatccttcg caagaccttc ctctatataa
9840
ggaagttcat ttcatttgga gaggacacgc tgaaatcacc agtctctctc tacaaatcta
9900
tctctctcga gatgattgaa caagatggat tgcacgcagg ttctccggcc gcttgggtgg
9960
agaggctatt cggctatgac tgggcacaac agacaatcgg ctgctctgat gccgccgtgt
10020
tccggctgtc agcgcagggg aggccggttc tttttgtcaa gaccgacctg tccggtgccc
10080
tgaatgaact tcaagacgag gcagcgcggc tatcgtggct ggccacgacg ggcgttcctt
10140
gcgcagctgt gctcgacgtt gtcactgaag cgggaaggga ctggctgcta ttgggcgaag
10200
tgccggggca ggatctcctg tcatctcacc ttgctcctgc cgagaaagta tccatcatgg
10260
ctgatgcaat gcggcggctg catacgcttg atccggctac ctgcccattc gaccaccaag
10320
cgaaacatcg catcgagcga gcacgtactc ggatggaagc cggtcttgtc gatcaggatg
10380
atctggacga agagcatcag gggctcgcgc cagccgaact gttcgccagg ctcaaggcgc
10440
gcatgcccga cggcgaggat ctcgtcgtga ctcatggcga tgcctgcttg ccgaatatca
10500
tggtggaaaa tggccgcttt tctggattca tcgactgtgg ccggctgggt gtggcggacc
10560
gctatcagga catagcgttg gctacccgtg atattgctga agagcttggc ggcgaatggg
10620
ctgaccgctt cctcgtgctt tacggtatcg ccgctcccga ttcgcagcgc atcgccttct
10680
atcgccttct tgacgagttc ttctgaaacg cgtgatcgtt caaacatttg gcaataaagt
10740
ttcttaagat tgaatcctgt tgccggtctt gcgatgatta tcatataatt tctgttgaat
10800
tacgttaagc atgtaataat taacatgtaa tgcatgacgt tatttatgag atgggttttt
10860
atgattagag tcccgcaatt atacatttaa tacgcgatag aaaacaaaat atagcgcgca
10920
aactaggata aattatcgcg cgcggtgtca tctatgttac tagatcgacg tccgtacggt
10980
taaaaccacc ccagtacatt aaaaacgtcc gcaatgtgtt attaagttgt ctaagcgtca
11040
atttgtttac accacaatat atcctgccac cagccagcca acagctcccc gaccggcagc
11100
tcggcacaaa atcaccactc gatacaggca gcccatcagt cc
11142
<210> 123
<211> 16749
<212> ДНК
<213> Искусственная последовательность
<220>
<223> последовательность вектора
<400> 123
gcttcctcgt gctttacggt atcgccgctc ccgattcgca gcgcatcgcc ttctatcgcc
60
ttcttgacga gttcttctga aacgcgtgat cgttcaaaca tttggcaata aagtttctta
120
agattgaatc ctgttgccgg tcttgcgatg attatcatat aatttctgtt gaattacgtt
180
aagcatgtaa taattaacat gtaatgcatg acgttattta tgagatgggt ttttatgatt
240
agagtcccgc aattatacat ttaatacgcg atagaaaaca aaatatagcg cgcaaactag
300
gataaattat cgcgcgcggt gtcatctatg ttactagatc gacgtccgta cggttaaaac
360
caccccagta cattaaaaac gtccgcaatg tgttattaag ttgtctaagc gtcaatttgt
420
ttacaccaca atatatcctg ccaccagcca gccaacagct ccccgaccgg cagctcggca
480
caaaatcacc actcgataca ggcagcccat cagtccacta gacgctcacc gggctggttg
540
ccctcgccgc tgggctggcg gccgtctatg gccctgcaaa cgcgccagaa acgccgtcga
600
agccgtgtgc gagacaccgc agccgccggc gttgtggata cctcgcggaa aacttggccc
660
tcactgacag atgaggggcg gacgttgaca cttgaggggc cgactcaccc ggcgcggcgt
720
tgacagatga ggggcaggct cgatttcggc cggcgacgtg gagctggcca gcctcgcaaa
780
tcggcgaaaa cgcctgattt tacgcgagtt tcccacagat gatgtggaca agcctgggga
840
taagtgccct gcggtattga cacttgaggg gcgcgactac tgacagatga ggggcgcgat
900
ccttgacact tgaggggcag agtgctgaca gatgaggggc gcacctattg acatttgagg
960
ggctgtccac aggcagaaaa tccagcattt gcaagggttt ccgcccgttt ttcggccacc
1020
gctaacctgt cttttaacct gcttttaaac caatatttat aaaccttgtt tttaaccagg
1080
gctgcgccct gtgcgcgtga ccgcgcacgc cgaagggggg tgccccccct tctcgaaccc
1140
tcccggcccg ctctcgcgtt ggcagcatca cccataattg tggtttcaaa atcggctccg
1200
tcgatactat gttatacgcc aactttgaaa acaactttga aaaagctgtt ttctggtatt
1260
taaggtttta gaatgcaagg aacagtgaat tggagttcgt cttgttataa ttagcttctt
1320
ggggtattta aatactgtag aaaagaggaa ggaaataata aatggctaaa atgagaatat
1380
caccggaatt gaaaaaactg atcgaaaaat accgctgcgt aaaagatacg gaaggaatgt
1440
ctcctgctaa ggtatataag ctggtgggag aaaatgaaaa cctatattta aaaatgacgg
1500
acagccggta taaagggacc acctatgatg tggaacggga aaaggacatg atgctatggc
1560
tggaaggaaa gctgcctgtt ccaaaggtcc tgcaccttga acggcatgat ggctggagca
1620
atctgctcat gagtgaggcc gatggcgtcc tttgctcgga agagtatgaa gatgaacaaa
1680
gccctgaaaa gattatcgag ctgtatgcgg agtgcatcag gctctttcac tccatcgaca
1740
tatcggattg tccctatacg aatagcttag acagccgctt agccgaattg gattacttac
1800
tgaataacga tctggccgat gtggattgcg aaaactggga agaagacacc ccatttaaag
1860
atccgcgcga gctgtatgat tttttaaaga cggaaaagcc cgaagaggaa cttgtctttt
1920
cccacggcga cctgggagac agcaacatct ttgtgaaaga tggcaaagta agtggcttta
1980
ttgatcttgg gagaagcggc agggcggaca agtggtatga cattgccttc tgcgtccggt
2040
cgatcaggga ggatattggg gaagaacagt atgtcgagct attttttgac ttactgggga
2100
tcaagcctga ttgggagaaa ataaaatatt atattttact ggatgaattg ttttagtacc
2160
tagatgtggc gcaacgatgc tggcgacaag caggagcgca ccgacttctt ccgcatcaag
2220
tgttttggct ctcaggccga ggcccacggc aagtatttgg gcaaggggtc gctggtattc
2280
gtgcagggca agattcggaa taccaagtac gagaaggacg gccagacggt ctacgggacc
2340
gacttcattg ccgataaggt ggattatctg gacaccaagg caccaggcgg atcaaatcag
2400
gaataagggc acattgcccc ggcgtgagtc ggggcaatcc cgcaaggagg gtgaatgaat
2460
cggacgtttg accggaaggc atacaggcaa gaactgatcg acgcggggtt ttccgccgag
2520
gatgccgaaa ccatcgcaag ccgcaccgtc atgcgtgcgc cccgcgaaac cttccagtcc
2580
gtcggctcga tggcccagca agctacggcc aagatcgagc gcgacagcgt gcaactggct
2640
ccccctgccc tgcccgcgcc atcggccgcc gtggagcgtt cgcgtcgtct cgaacaggag
2700
gcggcaggtt tggcgaagtc gatgaccatc gacacgcgag gaactatgac gaccaagaag
2760
cgaaaaaccg ccggcgagga cctggcaaaa caggtcagcg aggccaagca agccgcgttg
2820
ctgaaacaca cgaagcagca gatcaaggaa atgcagcttt ccttgttcga tattgcgccg
2880
tggccggaca cgatgcgagc gatgccaaac gacacggccc gctctgccct gttcaccacg
2940
cgcaacaaga aaatcccgcg cgaggcgctg caaaacaagg tcattttcca cgtcaacaag
3000
gacgtgaaga tcacctacac cggcgtcgag ctgcgggccg acgatgacga actggtgtgg
3060
cagcaggtgt tggagtacgc gaagcgcacc cctatcggcg agccgatcac cttcacgttc
3120
tacgagcttt gccaggacct gggctggtcg atcaatggcc ggtattacac gaaggccgag
3180
gaatgcctgt cgcgcctaca ggcgacggcg atgggcttca cgtccgaccg cgttgggcac
3240
ctggaatcgg tgtcgctgct gcaccgcttc cgcgtcctgg accgtggcaa gaaaacgtcc
3300
cgttgccagg tcctgatcga cgaggaaatc gtcgtgctgt ttgctggcga ccactacacg
3360
aaattcatat gggagaagta ccgcaagctg tcgccgacgg cccgacggat gttcgactat
3420
ttcagctcgc accgggagcc gtacccgctc aagctggaaa ccttccgcct catgtgcgga
3480
tcggattcca cccgcgtgaa gaagtggcgc gagcaggtcg gcgaagcctg cgaagagttg
3540
cgaggcagcg gcctggtgga acacgcctgg gtcaatgatg acctggtgca ttgcaaacgc
3600
tagggccttg tggggtcagt tccggctggg ggttcagcag ccagcgcttt actgagatcc
3660
tcttccgctt cctcgctcac tgactcgctg cgctcggtcg ttcggctgcg gcgagcggta
3720
tcagctcact caaaggcggt aatacggtta tccacagaat caggggataa cgcaggaaag
3780
aacatgtgag caaaaggcca gcaaaaggcc aggaaccgta aaaaggccgc gttgctggcg
3840
tttttccata ggctccgccc ccctgacgag catcacaaaa atcgacgctc aagtcagagg
3900
tggcgaaacc cgacaggact ataaagatac caggcgtttc cccctggaag ctccctcgtg
3960
cgctctcctg ttccgaccct gccgcttacc ggatacctgt ccgcctttct cccttcggga
4020
agcgtggcgc tttctcatag ctcacgctgt aggtatctca gttcggtgta ggtcgttcgc
4080
tccaagctgg gctgtgtgca cgaacccccc gttcagcccg accgctgcgc cttatccggt
4140
aactatcgtc ttgagtccaa cccggtaaga cacgacttat cgccactggc agcagccact
4200
ggtaacagga ttagcagagc gaggtatgta ggcggtgcta cagagttctt gaagtggtgg
4260
cctaactacg gctacactag aagaacagta tttggtatct gcgctctgct gaagccagtt
4320
accttcggaa aaagagttgg tagctcttga tccggcaaac aaaccaccgc tggtagcggt
4380
ggtttttttg tttgcaagca gcagattacg cgcagaaaaa aaggatctca agaagatcct
4440
ttgatctttt ctacggggtc tgacgctcag tggaacgaaa actcacgtta agggattttg
4500
gtcatgagat tatcaaaaag gatcttcacc tagatccttt tggatctcct gtggttggca
4560
tgcacataca aatggacgaa cggataaacc ttttcacgcc cttttaaata tccgattatt
4620
ctaataaacg ctcttttctc ttaggtttac ccgccaatat atcctgtcaa acactgatag
4680
tttaaactga aggcgggaaa cgacaatctg ctagtggatc tcccagtcac gacgttgtaa
4740
aacgggcgtc tgcgatcgct gaagttccta tacttttcag agaataggaa cttcggaata
4800
ggaacttccc atgggatcta gtaacataga tgacaccgcg cgcgataatt tatcctagtt
4860
tgcgcgctat attttgtttt ctatcgcgta ttaaatgtat aattgcggga ctctaatcat
4920
aaaaacccat ctcataaata acgtcatgca ttacatgtta attattacat gcttaacgta
4980
attcaacaga aattatatga taatcatcgc aagaccggca acaggattca atcttaagaa
5040
actttattgc caaatgtttg aacgatcacg ctagcggata acaatttcac acagggatat
5100
cactagtaaa aggtaccgag ctcctgcagt atcgatgcgg ccgcaaagtc gacgaattct
5160
cattagcaga actcaagatg ctgatcctct ggaacgttga acttgagctt gtgttcctcg
5220
aaaagcttgc acaactcttt gatgtaacgc tggtgaagtc tatcaacttc ctctctagaa
5280
ggctgaggag tcatttgaac ctcgataggc tttccaacga tagtagtgat aggctgtctg
5340
aaaggcatga gtccgaaaga gtattggaaa actccccttc catggaaaag tggaaggctg
5400
attcccataa tcttttggag tctgttctgg atccatctaa gccaagttcc aggagtgttc
5460
tcaacctggt tgaagaggtt gttctctccg aatgagaaga taggaacaag agcagcacca
5520
tgcataagag caagtctgat gaatccctta cggttcttca agagaagtct gtaagcacca
5580
ggtctagcat caagagcctc ttgagcacct ccaacgatga tagcaagaag gtttccacca
5640
ccctttctgc taaggatgtg atcagcagaa actttctcgc tagacacgag tccaccagac
5700
atgatgtaat ctctgaagaa tggagccctg aaccaaacgg taagcatcat aaggtaggat
5760
ctgattccag ggaacaaaga ggtgaatcca gtagactcag tacagaggtt aaggaaagca
5820
ccagcagcaa gaacaccatg aggatggaat ccagcaatgt agttacggct aggatcaagc
5880
tcagcagtct taacgagaga cacagggaag taatccttca tgtacttcca gatggccaat
5940
cttctgaaga attggatagg tctaccacct tgtctaggct tatcccaatc caagtaccac
6000
caggtagcgt aaagaacaga gaaaagccag aacctggtga acaagagtcc aacgaagata
6060
acgatgcaga gttgagcaag agcaaggaat gagaaaaccc actgaagaac agcgaaagtc
6120
tgcaatcttc tctcccaagg aacaagaagt ggagcgaact cgaccatgaa ttcagtcccc
6180
cgtgttctct ccaaatgaaa tgaacttcct tatatagagg aagggtcttg cgaaggatag
6240
tgggattgtg cgtcatccct tacgtcagtg gagatatcac atcaatccac ttgctttgaa
6300
gacgtggttg gaacgtcttc tttttccacg atgctcctcg tgggtggggg tccatctttg
6360
ggaccactgt cggcagaggc atcttcaacg atggcctttc ctttatcgca atgatggcat
6420
ttgtaggagc caccttcctt ttccactatc ttcacaataa agtgacagat agctgggcaa
6480
tggaatccga ggaggtttcc ggatattacc ctttgttgaa aagtctcaat tgccctttgg
6540
tcttctgaga ctgtatcttt gatatttttg gagtagacaa gtgtgtcgtg ctccaccatg
6600
ttgacgaaga ttttcttctt gtcattgagt cgtaagagac tctgtatgaa ctgttcgcca
6660
gtctttacgg cgagttctgt taggtcctct atttgaatct ttgactccat gggatccaag
6720
ggccctagaa tctaattatt ctattcagac taaattagta taagtatttt tttaatcaat
6780
aaataataat taataattta ttagtaggag tgattgaatt tataatatat tttttttaat
6840
catttaaaga atcttatatc tttaaattga caagagtttt aaatggggag agtgttatca
6900
tatcacaagt aggattaatg tgttatagtt tcacatgcat tacgataagt tgtgaaagat
6960
aacattatta tatataacaa tgacaatcac tagcgatcga gtagtgagag tcgtcttatt
7020
acactttctt ccttcgatct gtcacatggc ggcggcccga attctcacac aaggtagttg
7080
caagacactg aagtggtggt agtggtagta gaagaagcag aatcggtaga aaggcaagac
7140
aatggagaag atgaagatgg tggagattct cttcccacaa cgcagcaatc aaggttttca
7200
aggttaaggc actcgtgctt tccatcatcg aacatgaagt cgatgttatc ctcgaaagca
7260
agctcgttga agagttctgg gtactcaatt gggttctcgt tagcaaggtt ttgatcggta
7320
aggaatgggg agaatccagt atccatcatg cagaagttcc aagcaagttc gttgttatct
7380
ccgcacctat ccatttccat gatggtggaa gaatcaatgc agcagttaac aacggcagct
7440
tcctcagaat atcccacaat ttcagcctct tgttgctcag ccttctcttc ctctttttct
7500
tcttcctctt gaggtggttc ctcaacgtat tgttgcttaa cctcttccct aggttcctct
7560
ttagcttctc tagtctcaac ctcttgctta gcctcaacaa gaataccctc ttgatggtta
7620
gcctggttaa ctgggaatgg gaaaacgccc ttcttcttaa gcctgtcgat gtagttggag
7680
atatcgaagt tggtaacagc gttagcacct ctgtactcaa tagcagccat atcataagca
7740
gctgcagcct cttcttgagt gttgtaagtt ccgaggtaga ggtacttgtt tccgaaaact
7800
cttccaatcc tagcttccca tcttccgtta tgatgatgcc tagcaactcc cctatactta
7860
gaaactcccc tagagaatcc agatgactgc cttctaaggg aagcaagata ctcttctttg
7920
gtcaccctct gcatctcttc aagttctttg gtgtaagtct cagctgggaa gttaagaatg
7980
gtatctgggc cccaatactt aagagcagca agatcatagg tatgagcagc agcctcttca
8040
gaatcataag ctccaaggta aacctgcttg cccttcttgt tttggatgga gttccaagag
8100
gacttatccc aaaggtgagc ttcgaatctt ccagtccatc tatgcctagt aacacctctg
8160
tagatagatg accttctggt agaagctgga gaagttgggt tatgagactt atcgccagat
8220
ggagatgact tcttagccct cttagctctc tttggtcttg gagcttcaga ttgaattggg
8280
ctagaggtag tagtagaaga ggacactgaa gaagatggag aactagagca ggtagaggta
8340
gtgagcctct tcttcatgaa ttcactagtg attaaatttt gttggtgctt tgagcatata
8400
acaagcatgg tatatatagg cacgtaaaca agttgagaaa ttttactttg agtttgacat
8460
aaccaataaa agttagtgct gtttattacc tcactcagtt tgcaccgcaa ctgtcgttag
8520
tgatgtttac ctttcctttt tctattattt attagtatta tataatatat atatatgtgt
8580
gatgagactt gaaattgttt agcaccgcaa atgtccttct tgaggggagg ttttcttttg
8640
ctgaggttgg ggtgtcacat acacccccct ctatggactc aacgtccttg ctgaggttta
8700
ccccacacta catgagattt ttctagactc aatactatga tatttctcgc cttatcggaa
8760
ttggttaaac tcagttgaag ttagggtcat atcgataaaa ttgacacatg atcgactctg
8820
atattaaaca gattctctcc ctcgaacctc actcactttc ctttttctat tctttattag
8880
tattatataa tagatccgtt ccaaccattc acgtacataa gaagagagat attttttttt
8940
aatggactaa catgacaaat aaaacaaaca aaggagtaat gatcactaca acaaattaga
9000
ttatgaggga caaataattt catcatctat aaatcatgtt tcgtcactaa aaattttgtg
9060
tgacgaaaaa gatttcgtca atcagttgtc actaaaaata tacaaagacg atttaatgat
9120
gtttaccttt ccttttctat tctttattag tattatataa taaatatatg tgtgatgaga
9180
cttgaaattg tttagcaccg caaatgtcct tgttgaaggg aggttttctt ttgctgaggt
9240
tggggtgtca catacacccc ctctatggac tgaacgtcct ttttgaggtt tattttacac
9300
tgcatgagat ttttctagat tcaacattat gatttctaga ctcaacacta cgatcgtcac
9360
taaagactat tttttatata taaaaaaaat actttgtcct taaatgtata aattagggat
9420
aaatttatta ttataaaaaa ggttaataat tttgtgatta aatctattat tttgtcactg
9480
aaagtgtttg cttttaccga cgacatatat gtcactaaat attatcataa gtagtgacaa
9540
ttacaattgt cacaaaataa aaaaaattat tcatattcaa caaaaaaggg tactacgaca
9600
atacattttt tgtcactgaa agtaatcaag ttgtgataaa ttaatttatt taatgacaaa
9660
aatatttgta tcaaaattca cccatgatca tataataaaa ataactaaaa ttatactaaa
9720
gcataaatga caagaaaatc taactaaaac atatcaaata ttactcctaa acaaagacat
9780
ataagtaaaa atttcttcca aagtatcaat aacgtggtga cacatagctt gcaatcaatc
9840
ttgcttcaat tttcaccttt tatacctgta aaaagaaaga gaaaataaaa caatgattta
9900
aaaatcgaat tcccgcggcc cctagaatct aattattcta ttcagactaa attagtataa
9960
gtattttttt aatcaataaa taataattaa taatttatta gtaggagtga ttgaatttat
10020
aatatatttt ttttaatcat ttaaagaatc ttatatcttt aaattgacaa gagttttaaa
10080
tggggagagt gttatcatat cacaagtagg attaatgtgt tatagtttca catgcattac
10140
gataagttgt gaaagataac attattatat ataacaatga caatcactag cgatcgagta
10200
gtgagagtcg tcttattaca ctttcttcct tcgatctgtc acatggcggc ggcccgcggc
10260
cgcttcatta ctcgagccag gaggatggat cgatgctggt ctgagaccct gctaccggtt
10320
gctgactgaa ctgctcggca cggtccttca tttcacgggc cttgctcgcc aactttgtct
10380
tggccgactc caactgatcc gctccgggtg gatgtttccc cgtcaggtaa cggtagatcc
10440
aggacagcac agacagagcg gcaacaccaa atcccccgct tgccagaaaa cccgctccca
10500
acaggaagat ggtgatgact gcagatcaga aaaactcaga ttaatcgaca aattcgatcg
10560
cacaaactag aaactaacac cagatctaga tagaaatcac aaatcgaaga gtaattattc
10620
gacaaaactc aaattatttg aacaaatcgg atgatatcta tgaaacccta atcgagaatt
10680
aagatgatat ctaacgatca aacccagaaa atcgtcttcg atctaagatt aacagaatct
10740
aaaccaaaga acatatacga aattgggatc gaacgaaaac aaaatcgaag attttgagag
10800
aataaggaac acagaaattt acctgcaggg accagtacag gcgagaagat caccaggaga
10860
ggtgtggcga ttgtcagcgc aatgaccgtt ccagccaggg tcaacccgga taacaccaac
10920
aggctacctc cggcagtaac cgcggtcgct gcctttacaa cacgctgagc acgcggttgc
10980
agttgcaagt ggggggcacg tgtttgttgc tgctgcccgt agtgctctgc catggaaatt
11040
ttgttggtgc tttgagcata taacaagcat ggtatatata ggcacgtaaa caagttgaga
11100
aattttactt tgagtttgac ataaccaata aaagttagtg ctgtttatta cctcactcag
11160
tttgcaccgc aactgtcgtt agtgatgttt acctttcctt tttctattat ttattagtat
11220
tatataatat atatatatgt gtgatgagac ttgaaattgt ttagcaccgc aaatgtcctt
11280
cttgagggga ggttttcttt tgctgaggtt ggggtgtcac atacaccccc ctctatggac
11340
tcaacgtcct tgctgaggtt taccccacac tacatgagat ttttctagac tcaatactat
11400
gatatttctc gccttatcgg aattggttaa actcagttga agttagggtc atatcgataa
11460
aattgacaca tgatcgactc tgatattaaa cagattctct ccctcgaacc tcactcactt
11520
tcctttttct attctttatt agtattatat aatagatccg ttccaaccat tcacgtacat
11580
aagaagagag atattttttt ttaatggact aacatgacaa ataaaacaaa caaaggagta
11640
atgatcacta caacaaatta gattatgagg gacaaataat ttcatcatct ataaatcatg
11700
tttcgtcact aaaaattttg tgtgacgaaa aagatttcgt caatcagttg tcactaaaaa
11760
tatacaaaga cgatttaatg atgtttacct ttccttttct attctttatt agtattatat
11820
aataaatata tgtgtgatga gacttgaaat tgtttagcac cgcaaatgtc cttgttgaag
11880
ggaggttttc ttttgctgag gttggggtgt cacatacacc ccctctatgg actgaacgtc
11940
ctttttgagg tttattttac actgcatgag atttttctag attcaacatt atgatttcta
12000
gactcaacac tacgatcgtc actaaagact attttttata tataaaaaaa atactttgtc
12060
cttaaatgta taaattaggg ataaatttat tattataaaa aaggttaata attttgtgat
12120
taaatctatt attttgtcac tgaaagtgtt tgcttttacc gacgacatat atgtcactaa
12180
atattatcat aagtagtgac aattacaatt gtcacaaaat aaaaaaaatt attcatattc
12240
aacaaaaaag ggtactacga caatacattt tttgtcactg aaagtaatca agttgtgata
12300
aattaattta tttaatgaca aaaatatttg tatcaaaatt cacccatgat catataataa
12360
aaataactaa aattatacta aagcataaat gacaagaaaa tctaactaaa acatatcaaa
12420
tattactcct aaacaaagac atataagtaa aaatttcttc caaagtatca ataacgtggt
12480
gacacatagc ttgcaatcaa tcttgcttca attttcacct tttatacctg taaaaagaaa
12540
gagaaaataa aacaatgatt taaaggcgcg ccgcgtattg gctagagcag cttgccaaca
12600
tggtggagca cgacactctc gtctactcca agaatatcaa agatacagtc tcagaagacc
12660
aaagggctat tgagactttt caacaaaggg taatatcggg aaacctcctc ggattccatt
12720
gcccagctat ctgtcacttc atcaaaagga cagtagaaaa ggaaggtggc acctacaaat
12780
gccatcattg cgataaagga aaggctatcg ttcaagatgc ctctgccgac agtggtccca
12840
aagatggacc cccacccacg aggagcatcg tggaaaaaga agacgttcca accacgtctt
12900
caaagcaagt ggattgatgt gataacatgg tggagcacga cactctcgtc tactccaaga
12960
atatcaaaga tacagtctca gaagaccaaa gggctattga gacttttcaa caaagggtaa
13020
tatcgggaaa cctcctcgga ttccattgcc cagctatctg tcacttcatc aaaaggacag
13080
tagaaaagga aggtggcacc tacaaatgcc atcattgcga taaaggaaag gctatcgttc
13140
aagatgcctc tgccgacagt ggtcccaaag atggaccccc acccacgagg agcatcgtgg
13200
aaaaagaaga cgttccaacc acgtcttcaa agcaagtgga ttgatgtgat atctccactg
13260
acgtaaggga tgacgcacaa tcccactatc cttcgcaaga ccttcctcta tataaggaag
13320
ttcatttcat ttggagagga cacgctgaaa tcaccagtct ctctctacaa atctatctct
13380
gcgatcgcat ggcgattttg gattctgctg gcgttactac ggtgacggag aacggtggcg
13440
gagagttcgt cgatcttgat aggcttcgtc gacggaaatc gagatcggat tcttctaacg
13500
gacttcttct ctctggttcc gataataatt ctccttcgga tgatgttgga gctcccgccg
13560
acgttaggga tcggattgat tccgttgtta acgatgacgc tcagggaaca gccaatttgg
13620
ccggagataa taacggtggt ggcgataata acggtggtgg aagaggcggc ggagaaggaa
13680
gaggaaacgc cgatgctacg tttacgtatc gaccgtcggt tccagctcat cggagggcga
13740
gagagagtcc acttagctcc gacgcaatct tcaaacagag ccatgccgga ttattcaacc
13800
tctgtgtagt agttcttatt gctgtaaaca gtagactcat catcgaaaat cttatgaagt
13860
atggttggtt gatcagaacg gatttctggt ttagttcaag atcgctgcga gattggccgc
13920
ttttcatgtg ttgtatatcc ctttcgatct ttcctttggc tgcctttacg gttgagaaat
13980
tggtacttca gaaatacata tcagaacctg ttgtcatctt tcttcatatt attatcacca
14040
tgacagaggt tttgtatcca gtttacgtca ccctaaggtg tgattctgct tttttatcag
14100
gtgtcacttt gatgctcctc acttgcattg tgtggctaaa gttggtttct tatgctcata
14160
ctagctatga cataagatcc ctagccaatg cagctgataa ggccaatcct gaagtctcct
14220
actacgttag cttgaagagc ttggcatatt tcatggtcgc tcccacattg tgttatcagc
14280
caagttatcc acgttctgca tgtatacgga agggttgggt ggctcgtcaa tttgcaaaac
14340
tggtcatatt caccggattc atgggattta taatagaaca atatataaat cctattgtca
14400
ggaactcaaa gcatcctttg aaaggcgatc ttctatatgc tattgaaaga gtgttgaagc
14460
tttcagttcc aaatttatat gtgtggctct gcatgttcta ctgcttcttc cacctttggt
14520
taaacatatt ggcagagctt ctctgcttcg gggatcgtga attctacaaa gattggtgga
14580
atgcaaaaag tgtgggagat tactggagaa tgtggaatat gcctgttcat aaatggatgg
14640
ttcgacatat atacttcccg tgcttgcgca gcaagatacc aaagacactc gccattatca
14700
ttgctttcct agtctctgca gtctttcatg agctatgcat cgcagttcct tgtcgtctct
14760
tcaagctatg ggcttttctt gggattatgt ttcaggtgcc tttggtcttc atcacaaact
14820
atctacagga aaggtttggc tcaacggtgg ggaacatgat cttctggttc atcttctgca
14880
ttttcggaca accgatgtgt gtgcttcttt attaccacga cctgatgaac cgaaaaggat
14940
cgatgtcatg agcgatcgcg atcgttcaaa catttggcaa taaagtttct taagattgaa
15000
tcctgttgcc ggtcttgcga tgattatcat ataatttctg ttgaattacg ttaagcatgt
15060
aataattaac atgtaatgca tgacgttatt tatgagatgg gtttttatga ttagagtccc
15120
gcaattatac atttaatacg cgatagaaaa caaaatatag cgcgcaaact aggataaatt
15180
atcgcgcgcg gtgtcatcta tgttactaga tccctgcagg gcgtattggc tagagcagct
15240
tgccaacatg gtggagcacg acactctcgt ctactccaag aatatcaaag atacagtctc
15300
agaagaccaa agggctattg agacttttca acaaagggta atatcgggaa acctcctcgg
15360
attccattgc ccagctatct gtcacttcat caaaaggaca gtagaaaagg aaggtggcac
15420
ctacaaatgc catcattgcg ataaaggaaa ggctatcgtt caagatgcct ctgccgacag
15480
tggtcccaaa gatggacccc cacccacgag gagcatcgtg gaaaaagaag acgttccaac
15540
cacgtcttca aagcaagtgg attgatgtga taacatggtg gagcacgaca ctctcgtcta
15600
ctccaagaat atcaaagata cagtctcaga agaccaaagg gctattgaga cttttcaaca
15660
aagggtaata tcgggaaacc tcctcggatt ccattgccca gctatctgtc acttcatcaa
15720
aaggacagta gaaaaggaag gtggcaccta caaatgccat cattgcgata aaggaaaggc
15780
tatcgttcaa gatgcctctg ccgacagtgg tcccaaagat ggacccccac ccacgaggag
15840
catcgtggaa aaagaagacg ttccaaccac gtcttcaaag caagtggatt gatgtgatat
15900
ctccactgac gtaagggatg acgcacaatc ccactatcct tcgcaagacc ttcctctata
15960
taaggaagtt catttcattt ggagaggaca cgctgaaatc accagtctct ctctacaaat
16020
ctatctctct cgagatgatt gaacaagatg gattgcacgc aggttctccg gccgcttggg
16080
tggagaggct attcggctat gactgggcac aacagacaat cggctgctct gatgccgccg
16140
tgttccggct gtcagcgcag gggaggccgg ttctttttgt caagaccgac ctgtccggtg
16200
ccctgaatga acttcaagac gaggcagcgc ggctatcgtg gctggccacg acgggcgttc
16260
cttgcgcagc tgtgctcgac gttgtcactg aagcgggaag ggactggctg ctattgggcg
16320
aagtgccggg gcaggatctc ctgtcatctc accttgctcc tgccgagaaa gtatccatca
16380
tggctgatgc aatgcggcgg ctgcatacgc ttgatccggc tacctgccca ttcgaccacc
16440
aagcgaaaca tcgcatcgag cgagcacgta ctcggatgga agccggtctt gtcgatcagg
16500
atgatctgga cgaagagcat caggggctcg cgccagccga actgttcgcc aggctcaagg
16560
cgcgcatgcc cgacggcgag gatctcgtcg tgactcatgg cgatgcctgc ttgccgaata
16620
tcatggtgga aaatggccgc ttttctggat tcatcgactg tggccggctg ggtgtggcgg
16680
accgctatca ggacatagcg ttggctaccc gtgatattgc tgaagagctt ggcggcgaat
16740
gggctgacc
16749
<210> 124
<211> 137
<212> ДНК
<213> Искусственная последовательность
<220>
<223> последовательность линкера
<400> 124
atttaaatgc ggccgcgaat tcgtcgattg aggacgtccc tactagacct gctggacctc
60
ctcctgctac ttactacgat tctctcgctg tgcatatggt cagtcatgcc cgggcctgca
120
ggcggccgca tttaaat
137
<210> 125
<211> 434
<212> ДНК
<213> Искусственная последовательность
<220>
<223> шпРНКи
<400> 125
gtgagcaatg aaccaagatt tatcaatacc gttacttttg atagcaaaga gggttctcct
60
actcttgtta tggtccacgg atatggtgcc tctcagggtt tcttctttcg gaatttttat
120
gcccttgcga ggcatttcaa agttattgct attgatcagc ttggctgggg tggttcaagc
180
aggcctgact tcacatgcag aagtacagaa gagactgaag attggtttat tgattccttt
240
gaggagtggc gcaaagccaa aaaccttagc aactttattt tgcttgggca ctcctttgga
300
gggtatgtcg ctgcaaaata tgctctcaag catccagagc atgttcagca gttgattctg
360
gtaggaccag ctggatttac atcagagact gaacatatgt ccgagcggct tacccagttt
420
agagcaacat ggaa
434
<210> 126
<211> 593
<212> ДНК
<213> Искусственная последовательность
<220>
<223> шпРНКи
<400> 126
actgctgatg ctgtcaggca gtatctatgg ttgtttgagg agcataatgt tcttgaattc
60
ctcgtacttg ctggagatca tctatatcga atggattatg aaaagttcat tcaagcccac
120
agagaaacag atgctgatat tactgttgcc gcactgccaa tggatgaaaa gcgagccact
180
gcatttggtc tcatgaagat tgacgaagaa ggacgcatta ttgaatttgc agagaaaccg
240
aaaggagagc aattgaaagc aatgaaagtg gatactacca ttttaggtct tgatgatgag
300
agagctaaag agatgccttt tatcgcaagt atgggtatat atgtcattag caaagatgtg
360
atgttaaact tacttcgtga taagttccct ggtgccaatg attttggcag tgaagttatt
420
cctggtgcaa cttcgcttgg gatgagagtg caagcttatt tatatgatgg atactgggaa
480
gatattggta ccatcgaagc tttctacaat gccaatttgg gcattaccaa aaagccagtc
540
ccagatttta gcttctatga ccgatcagct ccaatctaca cccaacctcg ata
593
<210> 127
<211> 5
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> мотив липазы
<220>
<221> неопределенный признак
<222> (2)..(2)
<223> Xaa может представлять собой любую природную аминокислоту
<220>
<221> неопределенный признак
<222> (4)..(4)
<223> Xaa может представлять собой любую природную аминокислоту
<400> 127
Gly Xaa Ser Xaa Gly
1 5
<210> 128
<211> 6
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> мотив ацилтрансферазы
<220>
<221> X
<222> (2)..(5)
<223> любая аминокислота
<400> 128
His Xaa Xaa Xaa Xaa Asp
1 5
<210> 129
<211> 7
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> возможный связывающий липид мотив
<220>
<221> X
<222> (2)..(4)
<223> любая аминокислота
<400> 129
Val Xaa Xaa Xaa His Gly Phe
1 5
<210> 130
<211> 1224
<212> ДНК
<213> Arabidopsis thaliana
<400> 130
atggcggaag aaatctcaaa gacgaaggtg ggatcttctt ctactgcttc ggtggctgat
60
tcatctgctg ctgcgtcggc tgcaacgaat gcggccaaat caagatggaa aattttgtgg
120
cctaattcgc tccggtggat tcctacgtcc accgattaca tcatcgccgc cgagaaacgt
180
cttctctcca tcctcaagac gccttatgta caagagcaag tcagtattgg ttcaggacca
240
ccaggttcta aaatcaggtg gtttaggtct acgagcaatg agtcacgtta catcaacact
300
gttacatttg atgccaagga gggagctcct acactcgtca tggttcatgg ttatggtgct
360
tctcaagggt ttttcttccg taattttgat gctcttgcca gtcgatttag agtgatcgct
420
attgatcaac ttgggtgggg tggttcaagt aggcctgatt ttacatgtag aagcacagaa
480
gaaactgagg catggtttat cgactccttt gaggaatggc gtaaagccca gaatctcagt
540
aactttattc tattaggaca ttcttttgga ggctatgttg ctgctaaata cgcgcttaag
600
catcctgaac atgttcaaca cttaattctg gtgggatctg ctgggttctc agcagaagca
660
gatgccaaat cagaatggct cactaaattt agagcaacat ggaaaggtgc agtcctaaat
720
catttatggg agtcaaattt cactcctcag aagctggtta gaggattagg tccttggggt
780
ccaggtcttg taaatcggta tacaactgca agatttggtg cacattcgga gggaactggg
840
ctaacagaag aggaagccaa attgctaacc gattatgtgt accatacttt ggctgcaaag
900
gctagtggag agttatgctt gaaatacatc ttctcatttg gagcatttgc taggaagccc
960
ctcttacaaa gtgcatcaga gtggaaagtg ccaacaacgt ttatctatgg aatgaatgat
1020
tggatgaact atcaaggtgc ggtggaagcg aggaaatcca tgaaggtccc ttgcgaaatc
1080
attcgggttc cacagggtgg tcattttgtg ttcatagaca acccaattgg ttttcattct
1140
gcagtgcttt atgcttgccg caagtttata tctcaagact cctctcatga tcaacaactc
1200
ctagatggtc tacgattggt ttag
1224
<210> 131
<211> 1700
<212> ДНК
<213> Brachypodium distachyon
<400> 131
tccgcgcccg aaacgatccc aacagaagct ctaatctcca aagccgccgc gcgtgttgag
60
ggtggtgcgg ggaaaagctt ggtgtcgtga gcccccgtgt cgcatgcgcc gcgctgccgc
120
cgtgacgagg atggcagcga ccgaggagat gaggcaggcg tccgccgccg ccgccgccac
180
ggtgaccgag gcctcggcgt cggcggcccc gcccgcgggg tccaggtggg cgcgggtgtg
240
gccggccgcg ctgcgctgga tccccacctc caccgagcgc atcatcgccg ccgagaagcg
300
cctcctctcc gtactcaaaa ctgggtatgt ccaagaacaa gttaacattg gctcggctcc
360
acccgggtca aaagtaagat ggtttagatc atcaagtgat gagccaaggt tcatcaatac
420
agtgacattt gatagcaagg agaatgctcc cactcttgtc atggtccatg gttatggtgc
480
ttcacagggt ttcttcttta gaaattttga tgcccttgca agccgtttcc gagtgattgc
540
cattgatcag cttggttggg gtggatcaag tagacctgac ttcacctgta aaagtaccga
600
agaaactgag gcttggttca tagattcttt agaggaatgg cgtaaagcaa agaacctcag
660
taattttata ttgctcggtc attctttcgg aggatatgtt gcagcaaaat atgccttgca
720
gcatcctgaa cacgtgcagc acttaatttt ggtcggttct gctgggtttt catcagaaac
780
agatcatagc tctgagtggt taaccaagtt tcgagcaaca tggaaaggca tgctagtgaa
840
ccaactatgg gagtccaatt ttactcctca aagaattgta agaggattgg gtccttgggg
900
cccagatttg gttcgcagat ataccactgc taggtttggc tcatattcaa caggtgaatt
960
actaacagaa catgagtccg gcttgctgac agattacatt taccatacat tagctgccaa
1020
agctagtgga gagctgtgct tgaaatatat tttttccttg ggggcatttg caaggaaacc
1080
tcttctgcag agtgcatctg actggaaagt gccgaccact ttcatatatg gccatgacga
1140
ttggatgaaa taccaggggg cacagcaagc acgcaaggat atgaaagttc cttgcgaaat
1200
catcagagtc ccacagggag gacattttgt gttcatagat aacccttccg ggttccattc
1260
ggcagtcttc tatgcgtgcc ggaaattttt atctggagat gcagaggagg gtctctctct
1320
tcctgatggc ctgatatctg catgacagca tgaggcgcga tgtcatacca attagcggta
1380
tgaacacaaa gcaaagctat acggagctag gaaatgttac aaatgtcacg actcaccaga
1440
aatgttacaa atgtcaccac tcaccagttt cctttttgta tgtatgaatt gtgtgaatat
1500
acacgtcatt catatttgcc ggcgtatcag tacttcaata gtgataaaac atgatcatat
1560
atatatgtat gatttctcta gtcggttctc atcaagtcaa gttattgtga ttggtgaatg
1620
atatactttc caggtcaact ttgtgtttgc atgtacaaac tatcatggaa catatcagta
1680
tagtttatga tttgtcttcc
1700
<210> 132
<211> 1484
<212> ДНК
<213> Glycine max
<400> 132
gtcatggatg cgcgtcactg ctcgcgttca ttataatggc ggaagagata accaagaacg
60
acgtcggagt aacctccaaa accaccagaa gcagctccag gttctggcct cgttggattc
120
ccacttccac cgatcacatc atcgctgccg agaagcgcct tctttccgtc gtcaagactg
180
gttatgttca agagcatgtt aacattggct ctggtcctcc tggctccaaa gtgaggtggt
240
tccgttcatc cagcaacgag ccgcggttta ttaacaccgt tacatttgac agtaaacccc
300
attctccaac gcttgtcatg attcatggtt atgctgcttc acaggggttc ttttttcgca
360
attttgacgc gcttgcgtct cgatttagag tcattgctgt tgatcaactt ggatggggtg
420
gatcgagcag acctgatttc acatgcaaaa gcactgaaga aactgaggca tggtttattg
480
attcttttga ggaatggaga aaagccaaaa acttgagcaa ttttatactg ctcggacatt
540
cttttggtgg ttatgttgct gccaaatatg cgctcaagca ccctgagcat gtacaacact
600
tgattctggt tggatctgct ggattttcat ctgaatcaga tgcaaagtct gagtggataa
660
caaggtttcg agcaacatgg aagggggcag ttttgaacca tctttgggaa tcaaatttca
720
cacctcagaa acttgtcagg ggtttaggtc cttggggtcc caacatagtc cgcaagtata
780
caagtgctag gtttggtaca cattcaactg gggaaatact gactgaagag gaatcaacat
840
tgctgacaga ctatgtttac cacacattgg cggccaaagc tagtggagag ctgtgcttaa
900
aatatatttt ttcatttgga gcatttgcta ggatgcccct tcttctcagt gcctcagagt
960
ggaaggtgcc caccactttc atgtatggtt tccaagactg gatgaattat caaggtgccc
1020
aagaagctcg caagcatatg aaggttccat gcgaaatcat caggattcct cagggtgggc
1080
actttgcgtt cattgacaac ccaactgcct tccattcagc tgttttttat gcttgtcgaa
1140
ggtttcttac acctgatcca gacaatgaat ctcttcctaa agggctaacc tctgcatagg
1200
ttaggtctta attttgtgct attcctgtct atatgtattt taatattttt ttttactaat
1260
taaatttcat aattgaatga aatcatatgt atattgtttc agtaaagtgg aatttactga
1320
aaatatttgt aatagcaact tcaacaaaaa tcgatttgta ggagaaattt cttccctgga
1380
aattgttcta ttttaaatct tgttgctcat aagatattat gacttcattc aactaataat
1440
tcatgtcgtt taggaaaagt agttagttat attaaatttg tcaa
1484
<210> 133
<211> 1662
<212> ДНК
<213> Zea mays
<400> 133
accatacggg cggggccgca ccgaccgaac ctaaccgaga gcacgagcat acccgtcccg
60
actccgactg cagagcatca gccgaggaga aaagtcggga gaaacgcgcg tgacgtctgc
120
ccgcgtcgta tgcgccgcgc tgccgtcgcc gcgacgacga cgacgaccag gatggcagcc
180
gaggagatga gacgggcctc cgcctcaacg gccacggcgg agatgccggc gtcgccggcg
240
ccggcgcaag cggggtcgag gtgggcgcgg gtgtggccgc gcgcgctccg gtggatcccc
300
acctccacgg accgcatcat cgccgccgag aagcgactcc tcacgatagt caaaactgga
360
tatgtccagg aacgagtcaa cattggctct gctccacctg ggtcaaaagt aagatggttt
420
aggtcagcaa gtgatgaacc aaggttcatt aatactgtaa catttgatag caaggagaat
480
gcccccaccc tggttatggt ccatggctat ggagcttcac aggggttctt ctttcgaaac
540
tttgatgccc ttgcaagccg ttttagggtg attgccattg atcagcttgg ctggggtggt
600
tcaagcagac ctgacttcac atgtaaaagt accgaagaaa ctgaggcatg gttcatagat
660
tctttcgagg agtggcgcaa ggccaagaac ctcagtaatt ttatattgct tggtcactct
720
tttggaggat atgttgctgc aaaatatgcg ctaaagcacc ctgaacacgt tcaacagttg
780
attttggttg gtcctgctgg cttctcatca gaaacagagc atagctctga gtggttaacc
840
aagtttcgag caacatggaa aggcatgcta atgaatcgtc tttgggagtc caattttact
900
ccccaaaggg ttattagggg attgggtcct tggggtccag gtctagtaca gagatatacc
960
agtgccaggt ttggtacaag ttctactggt gaattactaa cagatgaaga atcggcattg
1020
atgacagatt atatgtacca tacgttagct gccaaagcta gtggagagct gtgcttgaaa
1080
tatatatttt ccttcggggc atttgcaagg aaacctcttc tgcagtgcgc gtccgattgg
1140
aaagtgccga ctactttcat atatgggcag caagattgga tgaactacca aggcgctcag
1200
caagcacgga aggacatgaa agttccttgt gaaataatca gggtgccgca gggtggacat
1260
tttgtgttca tagacaaccc ttcagggttc cactcggctg tcttctatgc gtgccgtaat
1320
cttctatcag taaatggaga ggagggattc acatttcctg atggcctaat atctgcgtga
1380
agtggcatgt tcaacaagct tgctcaacaa cagtttacat aaagcaaaga tatacgattg
1440
tggaaatcat tgcccatttc caccaatttg cttgtatacg gattatgctg tgtatatatt
1500
acataacaaa tgtattagta tcatttaatg cacgatttgt gaaagggcct gagtttgtat
1560
ttagcgaatt ttaggttggt ttttttccct ttttcttctt tcagtgcgct tgctagtcaa
1620
tcccatacta taagccgtga tcatttaaaa aaaaaaaaaa aa
1662
<210> 134
<211> 1763
<212> ДНК
<213> Sorghum bicolor
<400> 134
actgcagatg cgcggtcgtc ggctccggct cgcggaggcg agaacggcga accagcccgt
60
gtctctgttc cctttcttcc ctttaaaaac acggcaaaaa aaaagctagc cggttacgct
120
accgaaccga acggctcggc acgcgggcac gggcgcgggg tcgcaccgga aaagcacgag
180
cagagcagac ctgacgtctc cagactgcag gagcatcatc agtcgaggag gaggaagtgt
240
ggggggggga aagggaaacg tgcgcgtcgt atgcgcctcg ctgccgtcgc caggacgacc
300
aggatggcag ccgaggagat gaggcgggcc tcggcctccg cggcggtcgc ggccacgacg
360
gaggcggcgc cggcgccggc gcaagcgggg tccaggtggg cgcgggtgtg gccgagcgcg
420
ctccggtgga tccccacctc cacggatcgc atcatcgccg cggagaagcg gctcctctcg
480
atagtcaaaa ctgggtatgt ccaggaacaa gtcaacattg gctcagctcc acctgggtca
540
aaagtaagat ggtttaggtc agcgagtgat gaaccaaggt tcattaatac tgtaacattt
600
gatggcaagg agaacgcccc caccctggtt atggtccatg gctatggagc ttcacagggg
660
ttcttctttc gaaactttga cgcccttgca agtcgtttta gggtgattgc cattgatcag
720
cttggctggg gtggttcaag cagacctgac ttcacatgta aaagtaccga agaaactgag
780
gcatggttca tagattcttt tgaggagtgg cgcaaggcca agaacctcag taattttata
840
ttgcttggtc actcctttgg aggatatgtt gcggcaaagt atgccctaaa gcaccctgaa
900
cacattcagc acttagtttt ggttggtcct gctggcttct cgtcagaaac agaccatagc
960
tctgagtggt taaccaagtt tcgagcaaca tggaaaggca tgctagtgaa tcatctttgg
1020
gagtccaatt ttactcccca aagagttatt agaggattgg gcccttgggg tccaggtcta
1080
gtacaaagat ataccagtgc caggtttggt acacgttcaa ctggtgatat actaacagat
1140
caagaatcaa cattgttgac agattatatt taccatacct tagctgccaa agctagtgga
1200
gagctgtgct tgaaatatat attttccttc ggggcatttg caaggaaacc tcttctgcag
1260
tgcgcatccg attggaaagt gccgactact ttcatatatg gtcaggaaga ttggatgaac
1320
taccaagggg ctcagcaagc acggaaggac atgaaagttc cttgtgaaat aatcagggtg
1380
ccacagagtg gacattttgt gtttatagac aacccttcag ggttccactc ggctgtcttc
1440
tacgcgtgcc gtaatctttt atcccaaaat ggggaggagg gcttcacatt tcctggtggc
1500
ctaatatctg catgaagtgg catgttcaac aatcttatcg tgcccaacaa tagtttatat
1560
gaagcaaaga tatacgatgg tggaaatctt tgctcatttc caccaatctg gaaatatttg
1620
tgccctcttc caccaatttg tttgtatacg gattatgccg tgtatatatt ctgtgttgac
1680
tgtaagaaac ataatgtatt aacattatgt aatgtatgta cgattcttta tttgattttc
1740
aacttgcaat acgcaagaac cac
1763
<210> 135
<211> 1399
<212> ДНК
<213> Ricinus communis
<400> 135
cgccttttta ccagtcaatt tccattttta tatataagtg cttttgctta atttaagact
60
aactacagcg acgaattcgc gtttatgaaa ttgcttcgcc tacgactgct acgagtatct
120
agctcctcaa tatcatcaat aatggcggaa ggggctgctg ccacatcagc atcagcatca
180
gcgtcagcgt cagcgtcatg ggcaaaaaca agatctctac ggccatctgc tctccgttgg
240
atcccaactt caaccgatca catcatcgcc gccgaaaagc gtcttctctc cctcgtcaag
300
actccctatg ttgtggaaca agtgaatata gggtctggcc caccggggtc gaaggtgagg
360
tggtttcgtt ctaaaagcga cgaggcacgg tttattaaca cggttacttt tgatagcaaa
420
gaggaggatt ctcctacact ggttatggtt catggatatg ctgcttctca aggcttcttc
480
tttcgcaatt ttgatgctct tgcttctcgt ttcaggctca ttgctattga tcagctcggt
540
tggggtggat caagtagacc tgattttacg tgtaagagca ctgaagaaac tgaggcatgg
600
ttcattgact cctttgaggc ttggcgtaaa gagaaaaacc tcagtaactt catcttactt
660
ggacattctt tcggagggta tattgcagct aaatatgcac tcaagcatcc tgagcatgtt
720
caacatctga ttttagtggg atctgctgga ttttcatcag aatctgaaga caaatctgag
780
cagcttactc ggttcagagc aacatggaag ggagcagttt tgaatcattt atgggagtct
840
aattttactc ctcagaaggt tattagaggt ttaggtcctt ggggtccaga tctcgtacgc
900
aagtacacaa ctgctagatt tggttcatat tcaactggtg agatattaaa ggaggaggag
960
tccaaattgc ttacagacta tgtgtaccat accttagccg ccaaagctag tggagagcta
1020
tgcttgaaat atatattttc ttttggagca tttgctcgga tgccccttct acaaagcgcg
1080
tcacaatgga aagtgccaac tactttcata tatgggatgc aagattggat gaattatcaa
1140
ggggcccaaa gagctcgcaa agatatgaat gtcccatgtg aaatcattag ggttcctcag
1200
ggcgggcact tcgttttcat agacaaccca actgggtttc attcagctgt gttatatgcc
1260
tgccggagat ttctctcacc cgatcctgat aatgaatctc ttcctgaagg tctgatatct
1320
gcgtaggaag tgtggtttgt aattatttct tttttatttg ttgtgtataa tttatctgag
1380
aatttccaat tctttcaat
1399
<210> 136
<211> 1480
<212> ДНК
<213> Medicago truncatula
<400> 136
ggttggctca tagttccttt tacctgttga aaacaaaaca tatggagtaa cattttagtc
60
agaaattcaa agctacgcac ttgattaaac taattatcga aaaatggcgg aagaaattag
120
acaaaaggac gacgtcgatt catcttcgaa atctaaaagc ttctggtctt cactccgttg
180
gattcccact tctaccgatc atatcatcgc cgctgagaaa cgccttcttt ccattatcaa
240
gactgggtat gctcaagagc atgttaatat aggttctggt cctcctggct ctaaagttag
300
atggttccgt tcaaccagta acgagccacg ctttctcaac actgttacat ttgatagtaa
360
acccgattct cctacacttg ttatggttca tggatacgct gcttctcagg gtttcttctt
420
tcgcaatttt gatgctctcg cctctcgttt cagaatcatt gctgttgatc aacttggttg
480
gggaggatca agcagacctg atttcacatg caaaagtacc gaagaaactg aggcatggtt
540
cattgattct ttcgaggaat ggagaaaagc caaaaatctt accaatttca tactgcttgg
600
acattctttt ggtggttatg ttgcttccaa atacgcgctc aagcaccctc agcacgtaca
660
acacttaatt ttggtgggac ctgccgggtt tacagaagaa acagatccaa agactgagtt
720
tgttactaag tttcgagcaa catggaaggg agcagttctg aaccatctat gggaatctaa
780
ttttacacct cagaaaattg tcagaggttt aggtccttgg ggtcctaaca tggtccgcaa
840
atatacaagt gctaggtttg gtacacattc aaccgggcaa aaactgattg acgaggaatc
900
aagtctgctg actgattatg tttatcatac attggcggcc aaagctagtg gggagctgtg
960
tttaaaatat atttttgcat ttggagcatt tgctaggatg ccccttcttc aaagtgctca
1020
agagtggaag gtgcccacca cattcatata tggttacgaa gattggatga attatgaagg
1080
tgcccaagaa gctcgcaagc atatgaaggt tccatgtgaa attatcaggg tccctaaggc
1140
cggccatttt gtgttcattg acaacccaag tggcttccat tcagctgtgt tttatgcttg
1200
tcgaaggttt cttaccccaa attcggacaa tgaatctctt cccgaagggc tatcgtctgc
1260
ttaggattta attttgcatc aatccagtgt atattaatat ggttattaat ttttttttac
1320
ttcataactg aatgaagccg tgtcttgttt ctcagtgaag tggaatataa tggaaatata
1380
tgtaattgta ataacaataa tattgatttg ttggggaact ttgaggacaa aaacatattc
1440
tggtaaaatt ttgttgcaca tgcgacaaac atatgctgtg
1480
<210> 137
<211> 1317
<212> ДНК
<213> Arabidopsis thaliana
<400> 137
gatctctctc cctctctctc tctctctctc cgggaaaaat ggataacttc ttaccctttc
60
cctcttctaa cgcaaactct gtccaagaac tctctatgga tcctaacaac aatcgctcgc
120
acttcacaac agtccctact tatgatcatc atcaggctca gcctcatcac ttcttgcctc
180
cgttttcata cccggtggag cagatggcgg cggtgatgaa tcctcagccg gtttacttat
240
cggagtgtta tcctcagatc ccggttacgc aaaccggaag tgaattcggt tctctggttg
300
gtaatccttg tttgtggcaa gagagaggtg gttttcttga tccgcgtatg acgaagatgg
360
caaggatcaa caggaaaaac gccatgatga gatcaagaaa caactctagc cctaattcta
420
gtccaagtga gttggttgat tcaaagagac agctgatgat gcttaacttg aaaaataacg
480
tgcagatctc cgacaagaaa gatagctacc aacagtccac atttgataac aagaagctta
540
gggttttgtg tgagaaggaa ttgaagaaca gcgatgttgg gtcactcggg aggatagttc
600
taccaaagag agatgcagaa gcaaatcttc cgaagctatc tgataaagaa ggaatcgttg
660
tacagatgag agatgttttc tctatgcagt cttggtcttt caaatacaag ttttggtcca
720
ataacaagag cagaatgtat gtcctcgaga acacaggaga atttgtgaag caaaatggag
780
ctgagatagg agacttttta acaatatacg aggacgaaag caagaatctc tacttcgcca
840
tgaatggaaa ttcgggaaaa caaaatgaag gaagagaaaa tgagtcgagg gaaaggaacc
900
actacgaaga ggcaatgctt gattacatac caagagacga agaggaagct tccattgcaa
960
tgctcatcgg aaatctaaac gatcactatc ccatccctaa cgatctcatg gacctcacca
1020
ctgaccttca gcaccatcaa gccacgtcct catcaatgcc acctgaggat cacgcgtacg
1080
tgggttcatc cgatgatcag gtgagcttta acgactttga gtggtggtga tatggtggtg
1140
gaagttctca agttcataac cccctttatg aaaatagacc ttaagatata caaaagagat
1200
taaaagaaaa aaaagttagt atatttcatc atatctctca ttgaagatga gatttatatc
1260
tataattgtt taatagtgtt tttattactt ttctatcaat atattaaagt tttaatt
1317
<210> 138
<211> 1439
<212> ДНК
<213> Medicago truncatula
<400> 138
tttcatcctt acatattttg catattgaaa cacgtaggat ggaataagat tgataacaaa
60
aattgcattg tttgcatatt gaaaacatgg gacaattgca tgggttcatg tgcttcatta
120
taagccacac attaggaaac acaggttgat attcaccact atttaacata agaatatctc
180
atgtgtaagc attcatacaa atatcacaat tgaattaaaa accaaagaaa tgtcttcctc
240
taacttctct tgtatcctat ccatctcctt aacattcttc atcttgctac tgaacaaggt
300
gaattcagca gaaacaactt ccttttccat cacaaaattt gtcccagatc aaaagaatct
360
catcttccaa ggcgatgcga aaactgcctc aacagggaag ttagaactct ccaaggcagt
420
caagaactct attggtagag ctctttattc cgcccctatt cacatttggg atagcaaaac
480
cggtagtgtg gctaactttc aaactacctt cacctttaca ataacggcgc ctaatactta
540
taatgttgca gacggtcttg cattcttcat tgcaccaatt gatactaagc cgaaatcaat
600
tcatcatgga ggataccttg gagttttcga tagcaaaact tacaaaaaat caattcaaac
660
tgttgcagtt gaaattgaca ctttctataa tgctcaatgg gatccaaatc ccggaaatat
720
aagtagcact ggtcgacata ttggaatcga tgtaaactct atcaaatcaa taagcaccgt
780
gccgtggagt ttggaaaaca ataaaaaggc taatgttgcg atagggttta atggtgcaac
840
aaatgtgttg agtgttgatg tggaatatcc tttgattcgt cattataccc taagtcatgt
900
tgtgcctttg aaggatgttg ttcctgagtg ggtaaggatt ggtttctctt cttctactgg
960
agccgaatat tcagcacatg atattttatc gtggtctttt gattcaaagt tgaacctagg
1020
ttttgagaac aatatcaatg ccaatgtttc aagctctact caagctgcat agttgaaaac
1080
ttatccatta tgtatgtgtg agtgtaacca accagtctaa gaaaactata ataagatacc
1140
tgaaataatg gttcattatc gtgtagtaga aatatggtca caccatatct tctttttttt
1200
ttaataaatt atggaataat gctatttctc gcgagagtta tgtttcggaa agattcatga
1260
atagatgtta atcaattaga tctatatata tatatatata tatatatata tatatatata
1320
tagcattttc ttaaattatg catatgtaat atcgtgtaat gctattgttt atatcaatga
1380
atggtgtttt gtagtcacat aattcgtaat ttctctccat gagaacagcg aaccaatta
1439
<210> 139
<211> 1393
<212> ДНК
<213> Brassica napus
<400> 139
gagatgggta tccctataag gtgcagcatc gaaccatctg caacattttg actcgttttc
60
ttttgtgttt ttataacatc tgtctcttct tcactcgatc tccctctctt ttctttttca
120
atctccccaa cgaacctccc ttcataactc tctttctctc cccgggaaat atggataact
180
tcttgccctt ttcctcttct aacgcaaact ctgtccaaga actctccatg gatcttaaca
240
agaatcgctc gcacttctcc atggcgcagc ctcagcactt gttgccgcct tactcgtacg
300
ttgcatgtcc ggcacttgat cagacgggga ccatgaatca tcagcctctt cactcatcgg
360
atgcttttcc tcagatcccg gttgtacaaa ccggaggtga attcggctat ttggtttgta
420
agcccggtgt gaggcaggaa cgaggtggat ttcttgatcc acactccact aagatggcta
480
ggatcaacag gaagaaggcg atgctaagat caagaaacaa ctctaaccct aattctagtt
540
cgaatgagtt ggttgattca aggagacaag tggctcttac catgaaaaat aatgccgaga
600
ttgctgctag aaaagatttt tatcgattct cctcattcga taacaagaaa cttagggttt
660
tgttggtgaa gcacttgaag aacagcgatg ttgggtcact tggaaggatt gttctaccaa
720
agagagaagc agaaggaaat cttccggagc tatctgataa agaaggaatg gtattagaga
780
tgagagatgt tgactctgtg cagtcttggt ctttcaaata caagtactgg tccaataaca
840
agagcagaat gtatgtcctc gaaaacacag gagaatttgt gaagaaaaat ggagtattga
900
tgggagacta tctaacaatc tacgaggacg aaagcaagaa tctctacttc tccatcagaa
960
agcacccaca caaacaaaat gatggaagag aggatgagtc gatggaagtt atcgagatga
1020
acttctatga agatataatg tttgattaca taccaaatga tgaagacgat tccattgcaa
1080
tgctcctcgg aaatctaaac gagcactatc cctacccaaa tgatcttatg gatctcactg
1140
tcaatcttga tcagcatcag caagccacct cctcgtcgcc acctgctgat cacatgagct
1200
cgaacgattt cttatggtga tgtgatggac gttgatatgg attccctttg agatgatata
1260
caagggatga aaagaaaaga gtatcatatt catatccata tttgtttgat aaaatgtgtt
1320
tgttcccaat ctattattta tgaaaaactt atttgtgttt aactccagat taattaaata
1380
tttttcattt gac
1393
<210> 140
<211> 1755
<212> ДНК
<213> Arabidopsis thaliana
<400> 140
atgaactcga tgaataactg gttaggcttc tctctctctc ctcatgatca aaatcatcac
60
cgtacggatg ttgactcctc caccaccaga accgccgtag atgttgccgg agggtactgt
120
tttgatctgg ccgctccctc cgatgaatct tctgccgttc aaacatcttt tctttctcct
180
ttcggtgtca ccctcgaagc tttcaccaga gacaataata gtcactcccg agattgggac
240
atcaatggtg gtgcatgcaa taacattaac aataacgaac aaaatggacc aaagcttgag
300
aatttcctcg gccgcaccac cacgatttac aataccaacg agaccgttgt agatggaaat
360
ggcgattgtg gaggaggaga cggtggtggt ggcggctcac taggcctttc gatgataaaa
420
acatggctga gtaatcattc ggttgctaat gctaatcatc aagacaatgg taacggtgca
480
cgaggcttgt ccctctctat gaattcatct actagtgata gcaacaacta caacaacaat
540
gatgatgtcg tccaagagaa gactattgtt gatgtcgtag aaactacacc gaagaaaact
600
attgagagtt ttggacaaag gacgtctata taccgcggtg ttacaaggca tcggtggaca
660
ggtagatacg aggcacattt atgggacaat agttgcaaaa gagaaggcca gactcgcaaa
720
ggaagacaag tttatctggg aggttatgac aaagaagaaa aagcagctag ggcttacgat
780
ttagccgcac taaagtattg gggaaccacc actactacta acttcccctt gagtgaatat
840
gagaaagagg tagaagagat gaagcacatg acgaggcaag agtatgttgc ctctctgcgc
900
aggaaaagta gtggtttctc tcgtggtgca tcgatttatc gaggagtaac aaggcatcac
960
caacatggaa ggtggcaagc taggatcgga agagtcgccg gtaacaaaga cctctacttg
1020
ggaactttcg gcacacagga agaggctgct gaggcttatg acattgcagc cattaaattc
1080
agaggattaa gcgcagtgac taacttcgac atgaacagat acaatgttaa agcaatcctc
1140
gagagcccga gtctacctat tggtagttct gcgaaacgtc tcaaggacgt taataatccg
1200
gttccagcta tgatgattag taataacgtt tcagagagtg caaataatgt tagcggttgg
1260
caaaacactg cgtttcagca tcatcaggga atggatttga gcttattgca gcaacagcag
1320
gagaggtacg ttggttatta caatggagga aacttgtcta ccgagagtac tagggtttgt
1380
ttcaaacaag aggaggaaca acaacacttc ttgagaaact cgccgagtca catgactaat
1440
gttgatcatc atagctcgac ctctgatgat tctgttaccg tttgtggaaa tgttgttagt
1500
tatggtggtt atcaaggatt cgcaatccct gttggaacat cggttaatta cgatcccttt
1560
actgctgctg agattgctta caacgcaaga aatcattatt actatgctca gcatcagcaa
1620
caacagcaga ttcagcagtc gccgggagga gattttccgg tggcgatttc gaataaccat
1680
agctctaaca tgtactttca cggggaaggt ggtggagaag gggctccaac gttttcagtt
1740
tggaacgaca cttag
1755
<210> 141
<211> 2061
<212> ДНК
<213> Medicago truncatula
<400> 141
atgaacttgt taggtttctc tctatctcca caagaacaac atccatcaac acaagatcaa
60
acggtggctt cccgttttgg gttcaaccct aatgaaatct caggctctga tgttcaagga
120
gatcactgct atgatctctc ttctcacaca actcctcatc attcactcaa cctttctcat
180
cctttttcca tttatgaagc tttccacaca aataacaaca ttcacaccac tcaagattgg
240
aaggagaact acaacaacca aaacctacta ttgggaacat catgcatgaa ccaaaatgtg
300
aacaacaaca accaacaagc acaaccaaag ctagaaaact tcctcggtgg acactctttc
360
accgaccatc aagaatacgg tggtagcaac tcatactctt cattacacct cccacctcat
420
cagccggaag catcctgtgg cggtggtgat ggtagtacaa gtaacaataa ctcaataggt
480
ttatctatga taaaaacatg gctcagaaac caaccaccac caccagaaaa caacaacaat
540
aacaacaatg aaagtggtgc acgtgtgcag acactatcac tttctatgag tactggctca
600
cagtcaagtt catctgtgcc tcttctcaat gcaaatgtga tgagtggtga gatttcctca
660
tcggaaaaca aacaaccacc cacaactgca gttgtacttg atagcaacca aacaagtgtc
720
gttgaaagtg ctgtgcctag aaaatccgtt gatacatttg gacaaagaac ttccatttac
780
cgtggtgtaa caaggcatag atggacaggg agatatgaag ctcacctttg ggataatagt
840
tgtagaagag aggggcagac tcgcaaagga aggcaagttt acttgggagg ttatgacaaa
900
gaagaaaaag cagctagagc ctatgatttg gcagcactaa aatattgggg aacaactact
960
acaacaaatt ttccaattag ccattatgaa aaagaagtgg aagaaatgaa gcatatgaca
1020
aggcaagagt acgttgcgtc attgagaagg aaaagtagtg gtttttcacg aggtgcatcc
1080
atttaccgag gagtaacaag acatcatcaa catggtagat ggcaagctag gattggaaga
1140
gttgcaggca acaaagatct ctacctagga actttcagca ctcaagaaga ggcagcagag
1200
gcatatgatg tggcagcaat aaaattcaga ggactgagtg cagttacaaa ctttgacatg
1260
agcagatatg atgtcaaaac catacttgag agcagcacat taccaattgg tggtgctgca
1320
aagcgtttaa aagacatgga gcaagttgaa ttgaatcatg tgaatgttga tattagccat
1380
agaactgaac aagatcatag catcatcaac aacacttccc atttaacaga acaagccatc
1440
tatgcagcaa caaatgcatc taattggcat gcactttcat tccaacatca acaaccacat
1500
catcattaca atgccaacaa catgcagtta cagaattatc cttatggaac tcaaactcaa
1560
aagctttggt gcaaacaaga acaagattct gatgatcata gtacttatac tactgctact
1620
gatattcatc aactacagtt agggaataat aataacaata ctcacaattt ctttggttta
1680
caaaatatca tgagtatgga ttctgcttcc atggataata gttctggatc taattctgtt
1740
gtttatggtg gtggagatca tggtggttat ggaggaaatg gtggatatat gattccaatg
1800
gctattgcaa atgatggtaa ccaaaatcca agaagcaaca acaattttgg tgagagtgag
1860
attaaaggat ttggttatga aaatgttttt gggactacta ctgatcctta tcatgcacag
1920
gcagcaagga acttgtacta tcagccacaa caattatctg ttgatcaagg atcaaattgg
1980
gttccaactg ctattccaac acttgctcca aggactacca atgtctctct atgtcctcct
2040
ttcactttgt tgcatgaata g
2061
<210> 142
<211> 363
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 142
Met Asp Asn Phe Leu Pro Phe Pro Ser Ser Asn Ala Asn Ser Val Gln
1 5 10 15
Glu Leu Ser Met Asp Pro Asn Asn Asn Arg Ser His Phe Thr Thr Val
20 25 30
Pro Thr Tyr Asp His His Gln Ala Gln Pro His His Phe Leu Pro Pro
35 40 45
Phe Ser Tyr Pro Val Glu Gln Met Ala Ala Val Met Asn Pro Gln Pro
50 55 60
Val Tyr Leu Ser Glu Cys Tyr Pro Gln Ile Pro Val Thr Gln Thr Gly
65 70 75 80
Ser Glu Phe Gly Ser Leu Val Gly Asn Pro Cys Leu Trp Gln Glu Arg
85 90 95
Gly Gly Phe Leu Asp Pro Arg Met Thr Lys Met Ala Arg Ile Asn Arg
100 105 110
Lys Asn Ala Met Met Arg Ser Arg Asn Asn Ser Ser Pro Asn Ser Ser
115 120 125
Pro Ser Glu Leu Val Asp Ser Lys Arg Gln Leu Met Met Leu Asn Leu
130 135 140
Lys Asn Asn Val Gln Ile Ser Asp Lys Lys Asp Ser Tyr Gln Gln Ser
145 150 155 160
Thr Phe Asp Asn Lys Lys Leu Arg Val Leu Cys Glu Lys Glu Leu Lys
165 170 175
Asn Ser Asp Val Gly Ser Leu Gly Arg Ile Val Leu Pro Lys Arg Asp
180 185 190
Ala Glu Ala Asn Leu Pro Lys Leu Ser Asp Lys Glu Gly Ile Val Val
195 200 205
Gln Met Arg Asp Val Phe Ser Met Gln Ser Trp Ser Phe Lys Tyr Lys
210 215 220
Phe Trp Ser Asn Asn Lys Ser Arg Met Tyr Val Leu Glu Asn Thr Gly
225 230 235 240
Glu Phe Val Lys Gln Asn Gly Ala Glu Ile Gly Asp Phe Leu Thr Ile
245 250 255
Tyr Glu Asp Glu Ser Lys Asn Leu Tyr Phe Ala Met Asn Gly Asn Ser
260 265 270
Gly Lys Gln Asn Glu Gly Arg Glu Asn Glu Ser Arg Glu Arg Asn His
275 280 285
Tyr Glu Glu Ala Met Leu Asp Tyr Ile Pro Arg Asp Glu Glu Glu Ala
290 295 300
Ser Ile Ala Met Leu Ile Gly Asn Leu Asn Asp His Tyr Pro Ile Pro
305 310 315 320
Asn Asp Leu Met Asp Leu Thr Thr Asp Leu Gln His His Gln Ala Thr
325 330 335
Ser Ser Ser Met Pro Pro Glu Asp His Ala Tyr Val Gly Ser Ser Asp
340 345 350
Asp Gln Val Ser Phe Asn Asp Phe Glu Trp Trp
355 360
<210> 143
<211> 280
<212> БЕЛОК
<213> Medicago truncatula
<400> 143
Met Ser Ser Ser Asn Phe Ser Cys Ile Leu Ser Ile Ser Leu Thr Phe
1 5 10 15
Phe Ile Leu Leu Leu Asn Lys Val Asn Ser Ala Glu Thr Thr Ser Phe
20 25 30
Ser Ile Thr Lys Phe Val Pro Asp Gln Lys Asn Leu Ile Phe Gln Gly
35 40 45
Asp Ala Lys Thr Ala Ser Thr Gly Lys Leu Glu Leu Ser Lys Ala Val
50 55 60
Lys Asn Ser Ile Gly Arg Ala Leu Tyr Ser Ala Pro Ile His Ile Trp
65 70 75 80
Asp Ser Lys Thr Gly Ser Val Ala Asn Phe Gln Thr Thr Phe Thr Phe
85 90 95
Thr Ile Thr Ala Pro Asn Thr Tyr Asn Val Ala Asp Gly Leu Ala Phe
100 105 110
Phe Ile Ala Pro Ile Asp Thr Lys Pro Lys Ser Ile His His Gly Gly
115 120 125
Tyr Leu Gly Val Phe Asp Ser Lys Thr Tyr Lys Lys Ser Ile Gln Thr
130 135 140
Val Ala Val Glu Ile Asp Thr Phe Tyr Asn Ala Gln Trp Asp Pro Asn
145 150 155 160
Pro Gly Asn Ile Ser Ser Thr Gly Arg His Ile Gly Ile Asp Val Asn
165 170 175
Ser Ile Lys Ser Ile Ser Thr Val Pro Trp Ser Leu Glu Asn Asn Lys
180 185 190
Lys Ala Asn Val Ala Ile Gly Phe Asn Gly Ala Thr Asn Val Leu Ser
195 200 205
Val Asp Val Glu Tyr Pro Leu Ile Arg His Tyr Thr Leu Ser His Val
210 215 220
Val Pro Leu Lys Asp Val Val Pro Glu Trp Val Arg Ile Gly Phe Ser
225 230 235 240
Ser Ser Thr Gly Ala Glu Tyr Ser Ala His Asp Ile Leu Ser Trp Ser
245 250 255
Phe Asp Ser Lys Leu Asn Leu Gly Phe Glu Asn Asn Ile Asn Ala Asn
260 265 270
Val Ser Ser Ser Thr Gln Ala Ala
275 280
<210> 144
<211> 349
<212> БЕЛОК
<213> Brassica napus
<400> 144
Met Asp Asn Phe Leu Pro Phe Ser Ser Ser Asn Ala Asn Ser Val Gln
1 5 10 15
Glu Leu Ser Met Asp Leu Asn Lys Asn Arg Ser His Phe Ser Met Ala
20 25 30
Gln Pro Gln His Leu Leu Pro Pro Tyr Ser Tyr Val Ala Cys Pro Ala
35 40 45
Leu Asp Gln Thr Gly Thr Met Asn His Gln Pro Leu His Ser Ser Asp
50 55 60
Ala Phe Pro Gln Ile Pro Val Val Gln Thr Gly Gly Glu Phe Gly Tyr
65 70 75 80
Leu Val Cys Lys Pro Gly Val Arg Gln Glu Arg Gly Gly Phe Leu Asp
85 90 95
Pro His Ser Thr Lys Met Ala Arg Ile Asn Arg Lys Lys Ala Met Leu
100 105 110
Arg Ser Arg Asn Asn Ser Asn Pro Asn Ser Ser Ser Asn Glu Leu Val
115 120 125
Asp Ser Arg Arg Gln Val Ala Leu Thr Met Lys Asn Asn Ala Glu Ile
130 135 140
Ala Ala Arg Lys Asp Phe Tyr Arg Phe Ser Ser Phe Asp Asn Lys Lys
145 150 155 160
Leu Arg Val Leu Leu Val Lys His Leu Lys Asn Ser Asp Val Gly Ser
165 170 175
Leu Gly Arg Ile Val Leu Pro Lys Arg Glu Ala Glu Gly Asn Leu Pro
180 185 190
Glu Leu Ser Asp Lys Glu Gly Met Val Leu Glu Met Arg Asp Val Asp
195 200 205
Ser Val Gln Ser Trp Ser Phe Lys Tyr Lys Tyr Trp Ser Asn Asn Lys
210 215 220
Ser Arg Met Tyr Val Leu Glu Asn Thr Gly Glu Phe Val Lys Lys Asn
225 230 235 240
Gly Val Leu Met Gly Asp Tyr Leu Thr Ile Tyr Glu Asp Glu Ser Lys
245 250 255
Asn Leu Tyr Phe Ser Ile Arg Lys His Pro His Lys Gln Asn Asp Gly
260 265 270
Arg Glu Asp Glu Ser Met Glu Val Ile Glu Met Asn Phe Tyr Glu Asp
275 280 285
Ile Met Phe Asp Tyr Ile Pro Asn Asp Glu Asp Asp Ser Ile Ala Met
290 295 300
Leu Leu Gly Asn Leu Asn Glu His Tyr Pro Tyr Pro Asn Asp Leu Met
305 310 315 320
Asp Leu Thr Val Asn Leu Asp Gln His Gln Gln Ala Thr Ser Ser Ser
325 330 335
Pro Pro Ala Asp His Met Ser Ser Asn Asp Phe Leu Trp
340 345
<210> 145
<211> 584
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 145
Met Asn Ser Met Asn Asn Trp Leu Gly Phe Ser Leu Ser Pro His Asp
1 5 10 15
Gln Asn His His Arg Thr Asp Val Asp Ser Ser Thr Thr Arg Thr Ala
20 25 30
Val Asp Val Ala Gly Gly Tyr Cys Phe Asp Leu Ala Ala Pro Ser Asp
35 40 45
Glu Ser Ser Ala Val Gln Thr Ser Phe Leu Ser Pro Phe Gly Val Thr
50 55 60
Leu Glu Ala Phe Thr Arg Asp Asn Asn Ser His Ser Arg Asp Trp Asp
65 70 75 80
Ile Asn Gly Gly Ala Cys Asn Thr Leu Thr Asn Asn Glu Gln Asn Gly
85 90 95
Pro Lys Leu Glu Asn Phe Leu Gly Arg Thr Thr Thr Ile Tyr Asn Thr
100 105 110
Asn Glu Thr Val Val Asp Gly Asn Gly Asp Cys Gly Gly Gly Asp Gly
115 120 125
Gly Gly Gly Gly Ser Leu Gly Leu Ser Met Ile Lys Thr Trp Leu Ser
130 135 140
Asn His Ser Val Ala Asn Ala Asn His Gln Asp Asn Gly Asn Gly Ala
145 150 155 160
Arg Gly Leu Ser Leu Ser Met Asn Ser Ser Thr Ser Asp Ser Asn Asn
165 170 175
Tyr Asn Asn Asn Asp Asp Val Val Gln Glu Lys Thr Ile Val Asp Val
180 185 190
Val Glu Thr Thr Pro Lys Lys Thr Ile Glu Ser Phe Gly Gln Arg Thr
195 200 205
Ser Ile Tyr Arg Gly Val Thr Arg His Arg Trp Thr Gly Arg Tyr Glu
210 215 220
Ala His Leu Trp Asp Asn Ser Cys Lys Arg Glu Gly Gln Thr Arg Lys
225 230 235 240
Gly Arg Gln Val Tyr Leu Gly Gly Tyr Asp Lys Glu Glu Lys Ala Ala
245 250 255
Arg Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Pro Thr Thr Thr
260 265 270
Thr Asn Phe Pro Leu Ser Glu Tyr Glu Lys Glu Val Glu Glu Met Lys
275 280 285
His Met Thr Arg Gln Glu Tyr Val Ala Ser Leu Arg Arg Lys Ser Ser
290 295 300
Gly Phe Ser Arg Gly Ala Ser Ile Tyr Arg Gly Val Thr Arg His His
305 310 315 320
Gln His Gly Arg Trp Gln Ala Arg Ile Gly Arg Val Ala Gly Asn Lys
325 330 335
Asp Leu Tyr Leu Gly Thr Phe Gly Thr Gln Glu Glu Ala Ala Glu Ala
340 345 350
Tyr Asp Ile Ala Ala Ile Lys Phe Arg Gly Leu Ser Ala Val Thr Asn
355 360 365
Phe Asp Met Asn Arg Tyr Asn Val Lys Ala Ile Leu Glu Ser Pro Ser
370 375 380
Leu Pro Ile Gly Ser Ser Ala Lys Arg Leu Lys Asp Val Asn Asn Pro
385 390 395 400
Val Pro Ala Met Met Ile Ser Asn Asn Val Ser Glu Ser Ala Asn Asn
405 410 415
Val Ser Gly Trp Gln Asn Thr Ala Phe Gln His His Gln Gly Met Asp
420 425 430
Leu Ser Leu Leu Gln Gln Gln Gln Glu Arg Tyr Val Gly Tyr Tyr Asn
435 440 445
Gly Gly Asn Leu Ser Thr Glu Ser Thr Arg Val Cys Phe Lys Gln Glu
450 455 460
Glu Glu Gln Gln His Phe Leu Arg Asn Ser Pro Ser His Met Thr Asn
465 470 475 480
Val Asp His His Ser Ser Thr Ser Asp Asp Ser Val Thr Val Cys Gly
485 490 495
Asn Val Val Ser Tyr Gly Gly Tyr Gln Gly Phe Ala Ile Pro Val Gly
500 505 510
Thr Ser Val Asn Tyr Asp Pro Phe Thr Ala Ala Glu Ile Ala Tyr Asn
515 520 525
Ala Arg Asn His Tyr Tyr Tyr Ala Gln His Gln Gln Gln Gln Gln Ile
530 535 540
Gln Gln Ser Pro Gly Gly Asp Phe Pro Val Ala Ile Ser Asn Asn His
545 550 555 560
Ser Ser Asn Met Tyr Phe His Gly Glu Gly Gly Gly Glu Gly Ala Pro
565 570 575
Thr Phe Ser Val Trp Asn Asp Thr
580
<210> 146
<211> 686
<212> БЕЛОК
<213> Medicago truncatula
<400> 146
Met Asn Leu Leu Gly Phe Ser Leu Ser Pro Gln Glu Gln His Pro Ser
1 5 10 15
Thr Gln Asp Gln Thr Val Ala Ser Arg Phe Gly Phe Asn Pro Asn Glu
20 25 30
Ile Ser Gly Ser Asp Val Gln Gly Asp His Cys Tyr Asp Leu Ser Ser
35 40 45
His Thr Thr Pro His His Ser Leu Asn Leu Ser His Pro Phe Ser Ile
50 55 60
Tyr Glu Ala Phe His Thr Asn Asn Asn Ile His Thr Thr Gln Asp Trp
65 70 75 80
Lys Glu Asn Tyr Asn Asn Gln Asn Leu Leu Leu Gly Thr Ser Cys Met
85 90 95
Asn Gln Asn Val Asn Asn Asn Asn Gln Gln Ala Gln Pro Lys Leu Glu
100 105 110
Asn Phe Leu Gly Gly His Ser Phe Thr Asp His Gln Glu Tyr Gly Gly
115 120 125
Ser Asn Ser Tyr Ser Ser Leu His Leu Pro Pro His Gln Pro Glu Ala
130 135 140
Ser Cys Gly Gly Gly Asp Gly Ser Thr Ser Asn Asn Asn Ser Ile Gly
145 150 155 160
Leu Ser Met Ile Lys Thr Trp Leu Arg Asn Gln Pro Pro Pro Pro Glu
165 170 175
Asn Asn Asn Asn Asn Asn Asn Glu Ser Gly Ala Arg Val Gln Thr Leu
180 185 190
Ser Leu Ser Met Ser Thr Gly Ser Gln Ser Ser Ser Ser Val Pro Leu
195 200 205
Leu Asn Ala Asn Val Met Ser Gly Glu Ile Ser Ser Ser Glu Asn Lys
210 215 220
Gln Pro Pro Thr Thr Ala Val Val Leu Asp Ser Asn Gln Thr Ser Val
225 230 235 240
Val Glu Ser Ala Val Pro Arg Lys Ser Val Asp Thr Phe Gly Gln Arg
245 250 255
Thr Ser Ile Tyr Arg Gly Val Thr Arg His Arg Trp Thr Gly Arg Tyr
260 265 270
Glu Ala His Leu Trp Asp Asn Ser Cys Arg Arg Glu Gly Gln Thr Arg
275 280 285
Lys Gly Arg Gln Val Tyr Leu Gly Gly Tyr Asp Lys Glu Glu Lys Ala
290 295 300
Ala Arg Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Thr Thr Thr
305 310 315 320
Thr Thr Asn Phe Pro Ile Ser His Tyr Glu Lys Glu Val Glu Glu Met
325 330 335
Lys His Met Thr Arg Gln Glu Tyr Val Ala Ser Leu Arg Arg Lys Ser
340 345 350
Ser Gly Phe Ser Arg Gly Ala Ser Ile Tyr Arg Gly Val Thr Arg His
355 360 365
His Gln His Gly Arg Trp Gln Ala Arg Ile Gly Arg Val Ala Gly Asn
370 375 380
Lys Asp Leu Tyr Leu Gly Thr Phe Ser Thr Gln Glu Glu Ala Ala Glu
385 390 395 400
Ala Tyr Asp Val Ala Ala Ile Lys Phe Arg Gly Leu Ser Ala Val Thr
405 410 415
Asn Phe Asp Met Ser Arg Tyr Asp Val Lys Thr Ile Leu Glu Ser Ser
420 425 430
Thr Leu Pro Ile Gly Gly Ala Ala Lys Arg Leu Lys Asp Met Glu Gln
435 440 445
Val Glu Leu Asn His Val Asn Val Asp Ile Ser His Arg Thr Glu Gln
450 455 460
Asp His Ser Ile Ile Asn Asn Thr Ser His Leu Thr Glu Gln Ala Ile
465 470 475 480
Tyr Ala Ala Thr Asn Ala Ser Asn Trp His Ala Leu Ser Phe Gln His
485 490 495
Gln Gln Pro His His His Tyr Asn Ala Asn Asn Met Gln Leu Gln Asn
500 505 510
Tyr Pro Tyr Gly Thr Gln Thr Gln Lys Leu Trp Cys Lys Gln Glu Gln
515 520 525
Asp Ser Asp Asp His Ser Thr Tyr Thr Thr Ala Thr Asp Ile His Gln
530 535 540
Leu Gln Leu Gly Asn Asn Asn Asn Asn Thr His Asn Phe Phe Gly Leu
545 550 555 560
Gln Asn Ile Met Ser Met Asp Ser Ala Ser Met Asp Asn Ser Ser Gly
565 570 575
Ser Asn Ser Val Val Tyr Gly Gly Gly Asp His Gly Gly Tyr Gly Gly
580 585 590
Asn Gly Gly Tyr Met Ile Pro Met Ala Ile Ala Asn Asp Gly Asn Gln
595 600 605
Asn Pro Arg Ser Asn Asn Asn Phe Gly Glu Ser Glu Ile Lys Gly Phe
610 615 620
Gly Tyr Glu Asn Val Phe Gly Thr Thr Thr Asp Pro Tyr His Ala Gln
625 630 635 640
Ala Ala Arg Asn Leu Tyr Tyr Gln Pro Gln Gln Leu Ser Val Asp Gln
645 650 655
Gly Ser Asn Trp Val Pro Thr Ala Ile Pro Thr Leu Ala Pro Arg Thr
660 665 670
Thr Asn Val Ser Leu Cys Pro Pro Phe Thr Leu Leu His Glu
675 680 685
<210> 147
<211> 336
<212> ДНК
<213> Искусственная последовательность
<220>
<223> индуцибельный промотор
<400> 147
tcgatagttg tgatagttcc cacttgtccg tccgcatcgg catccgcagc tcgggatagt
60
tccgacctag gattggatgc atgcggaacc gcacgagggc ggggcggaaa ttgacacacc
120
actcctctcc acgcaccgtt caagaggtac gcgtatagag ccgtatagag cagagacgga
180
gcactttctg gtactgtccg cacgggatgt ccgcacggag agccacaaac gagcggggcc
240
ccgtacgtgc tctcctaccc caggatcgca tccccgcata gctgaacatc tatataaaga
300
cccccaaggt tctcagtctc accaacatca tcaacc
336
<210> 148
<211> 2466
<212> ДНК
<213> Искусственная последовательность
<220>
<223> индуктор
<400> 148
atggccgaca ctagaagaag gcagaaccac tcttgtgacc catgccgtaa gggcaagaga
60
agatgtgatg ctccagagaa ccgtaacgag gctaatgaga acggatgggt gtcatgctct
120
aactgcaaga ggtggaacaa ggactgcacc ttcaactggc ttagctccca aaggtctaag
180
gctaagggtg ctgctccaag agctaggact aagaaggcta ggactgctac tactacctcc
240
gagccttcta cttccgctgc tactattcca actcccgagt ccgataatca cgatgctcca
300
ccagtgatca actcccacga tgctttgcca tcttggactc agggacttct ttctcaccct
360
ggcgatctct tcgacttctc ccattctgct attccagcta acgctgagga tgctgctaac
420
gtgcaatctg atgctccatt cccatgggat cttgctatcc caggcgattt ctctatggga
480
cagcaacttg agaagcccct ctccccattg tctttccagg ctgttcttct tccaccacac
540
tccccaaaca ctgatgatct cattcgtgag cttgaggaac agactaccga tccagattcc
600
gtgactgaca ctaactccgt tcagcaagtt gctcaggatg gctctctttg gtctgatagg
660
cagtctccac tcctcccaga aaacagtttg tgcatggctt ccgactctac cgctagaagg
720
tatgctaggt ccaccatgac caagaacctc atgaggatct accacgactc catggaaaac
780
gccctttctt gctggcttac tgagcacaac tgcccatact ccgaccagat ttcttacctc
840
ccaccaaagc aaagggctga gtggggacca aattggtcta acaggatgtg cattagggtg
900
tgcaggctcg atagggtgtc aacttctctt agaggaaggg ctctctccgc tgaagaagat
960
aaggctgctg ctagggcact tcaccttgct attgtggctt tcgcttctca gtggactcaa
1020
catgctcaaa ggggagctgg acttaacgtc ccagctgata ttgctgctga cgagcgttct
1080
attaggcgta acgcttggaa tgaggctagg catgcacttc agcacactac tggaatccca
1140
tccttcaggg tgatcttcgc caacatcatc ttcagcctca ctcagtccgt gctcgatgat
1200
gatgagcaac atggaatggg agctaggctc gataagcttc tcgagaatga tggtgctcca
1260
gtgttcctcg agactgctaa taggcagctc tacaccttca ggcacaagtt cgctaggatg
1320
cagagaaggg gtaaggcttt caataggctt cctggtggat ccgtggcttc tactttcgct
1380
ggaattttcg agactcccac cccctcatct gagtctccac aacttgatcc agtggtggct
1440
tctgaggaac acaggtctac tctgtctctc atgttctggc tcgggatcat gttcgacact
1500
ctgtctgctg ctatgtacca gaggccactt gttgtgtccg atgaggactc ccagatctct
1560
tctgcttctc caccaagaag aggtgccgag actcctatta accttgattg ctgggagcca
1620
ccaaggcagg tcccatctaa tcaagagaag tctgatgtgt ggggcgacct gttccttagg
1680
acttctgatt ctttgcccga ccacgagtcc cacactcaaa tttctcaacc agctgctagg
1740
tggccatgca cttatgaaca agctgctgct gctctctcct ctgctactcc tgttaaggtg
1800
ttgctttaca ggcgtgtgac tcagctccag actttgttgt ataggggagc ttctccagct
1860
aggcttgagg ctgctattca gaggactctc tacgtgtaca accactggac tgctaagtac
1920
cagccattca tgcaggattg cgttgccaac catgagcttc tcccatccag gatccagtct
1980
tggtacgtga tccttgatgg acactggcac cttgctgcta tgcttttggc tgatgtgctc
2040
gagtccatcg acagggattc ctactccgat atcaaccaca tcgacctcgt gactaagctc
2100
aggcttgata acgctcttgc tgtgtctgct ctcgctaggt catctcttag aggccaagaa
2160
ctcgatccag gcaaggcttc tccaatgtac aggcacttcc acgactccct tactgaggtt
2220
gcattccttg ttgagccatg gactgtggtg ctcatccact catttgctaa ggctgcttac
2280
atcctcctcg attgccttga tcttgatggt cagggaaacg ctctcgctgg ataccttcaa
2340
cttaggcaga actgcaacta ctgcatcagg gctctccagt tccttggccg taagtctgat
2400
atggctgctc tcgtggctaa ggatcttgag aggggactca acggaaaggt cgacagcttc
2460
ctctaa
2466
<210> 149
<211> 208
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 149
Met Thr Ser Ser Val Ile Val Ala Gly Ala Gly Asp Lys Asn Asn Gly
1 5 10 15
Ile Val Val Gln Gln Gln Pro Pro Cys Val Ala Arg Glu Gln Asp Gln
20 25 30
Tyr Met Pro Ile Ala Asn Val Ile Arg Ile Met Arg Lys Thr Leu Pro
35 40 45
Ser His Ala Lys Ile Ser Asp Asp Ala Lys Glu Thr Ile Gln Glu Cys
50 55 60
Val Ser Glu Tyr Ile Ser Phe Val Thr Gly Glu Ala Asn Glu Arg Cys
65 70 75 80
Gln Arg Glu Gln Arg Lys Thr Ile Thr Ala Glu Asp Ile Leu Trp Ala
85 90 95
Met Ser Lys Leu Gly Phe Asp Asn Tyr Val Asp Pro Leu Thr Val Phe
100 105 110
Ile Asn Arg Tyr Arg Glu Ile Glu Thr Asp Arg Gly Ser Ala Leu Arg
115 120 125
Gly Glu Pro Pro Ser Leu Arg Gln Thr Tyr Gly Gly Asn Gly Ile Gly
130 135 140
Phe His Gly Pro Ser His Gly Leu Pro Pro Pro Gly Pro Tyr Gly Tyr
145 150 155 160
Gly Met Leu Asp Gln Ser Met Val Met Gly Gly Gly Arg Tyr Tyr Gln
165 170 175
Asn Gly Ser Ser Gly Gln Asp Glu Ser Ser Val Gly Gly Gly Ser Ser
180 185 190
Ser Ser Ile Asn Gly Met Pro Ala Phe Asp His Tyr Gly Gln Tyr Lys
195 200 205
<210> 150
<211> 236
<212> БЕЛОК
<213> Arabidopsis lyrata
<400> 150
Met Glu Arg Gly Ala Pro Phe Ser His Tyr Gln Leu Pro Lys Ser Ile
1 5 10 15
Ser Glu Leu Asn Leu Asp Gln His Ser Asn Pro Asn Pro Met Thr Ser
20 25 30
Ser Val Val Val Ala Asp Ala Ser Asp Asn Asn Lys Gly Ile Val Ala
35 40 45
Gln Gln Gln Pro Pro Cys Met Ala Arg Glu Gln Asp Gln Tyr Met Pro
50 55 60
Ile Ala Asn Val Ile Arg Ile Met Arg Lys Ile Leu Pro Ser His Ala
65 70 75 80
Lys Ile Ser Asp Asp Ala Lys Glu Thr Ile Gln Glu Cys Val Ser Glu
85 90 95
Tyr Ile Ser Phe Val Thr Gly Glu Ala Asn Glu Arg Cys Gln Arg Glu
100 105 110
Gln Arg Lys Thr Ile Thr Ala Glu Asp Ile Leu Trp Ala Met Ser Lys
115 120 125
Leu Gly Phe Asp Asn Tyr Val Asp Pro Leu Thr Val Phe Ile Asn Arg
130 135 140
Tyr Arg Glu Ile Glu Thr Asp Arg Gly Ser Ala Leu Arg Glu Pro Pro
145 150 155 160
Ser Leu Arg Gln Ala Tyr Gly Gly Asn Gly Ile Gly Phe His Gly Pro
165 170 175
Ser His Gly Leu Pro Pro Pro Gly Pro Tyr Gly Tyr Gly Met Leu Asp
180 185 190
Gln Ser Met Val Met Gly Gly Gly Arg Tyr Tyr Gln Asn Gly Ser Ser
195 200 205
Gly Gln Asp Glu Ser Ser Ala Gly Gly Gly Ser Ser Ser Ser Ile Asn
210 215 220
Gly Met Pro Ala Phe Asp Ser Tyr Gly Gln Tyr Lys
225 230 235
<210> 151
<211> 230
<212> БЕЛОК
<213> Brassica napus
<400> 151
Met Glu Arg Gly Ala Pro Leu Ser His Tyr Gln Leu Pro Lys Ser Asn
1 5 10 15
Ser Gly Leu Asn Leu Asp Gln His Asn Asn Ser Ile Pro Thr Met Thr
20 25 30
Gly Ser Ile Ser Ala Cys Asp Asp Lys Asn Lys Thr Ile Leu Pro Gln
35 40 45
Gln Gln Pro Ser Met Pro Arg Glu Gln Asp Gln Tyr Met Pro Ile Ala
50 55 60
Asn Val Ile Arg Ile Met Arg Lys Ile Leu Pro Pro His Ala Lys Ile
65 70 75 80
Ser Asp Asp Ala Lys Glu Thr Ile Gln Glu Cys Val Ser Glu Tyr Ile
85 90 95
Ser Phe Val Thr Gly Glu Ala Asn Glu Arg Cys Gln Arg Glu Gln Arg
100 105 110
Lys Thr Ile Thr Ala Glu Asp Ile Leu Trp Ala Met Ser Lys Leu Gly
115 120 125
Phe Asp Asp Tyr Val Gly Pro Leu Asn Val Phe Ile Asn Arg Tyr Arg
130 135 140
Glu Phe Glu Thr Asp Arg Gly Cys Ser Leu Arg Gly Glu Ser Ser Phe
145 150 155 160
Lys Pro Val Tyr Gly Gly Ser Gly Met Gly Phe His Gly Pro Pro Pro
165 170 175
Pro Gly Ser Tyr Gly Tyr Gly Met Leu Asp Gln Ser Met Val Met Gly
180 185 190
Gly Gly Arg Tyr Tyr His Asn Gly Ser Gly Gln Asp Gly Ser Val Ser
195 200 205
Gly Gly Gly Gly Ser Ser Ser Ser Met Asn Gly Met Pro Val Tyr Asp
210 215 220
Gln Tyr Gly Gln Tyr Lys
225 230
<210> 152
<211> 252
<212> БЕЛОК
<213> Ricinus communis
<400> 152
Met Glu Arg Gly Gly Arg Val His Arg Tyr Arg Arg His Ala Lys Gln
1 5 10 15
Pro Thr Pro Thr Thr Ser Ala Thr Ala Ser Thr Ser Pro Gly Met Ser
20 25 30
Ser Val Gln Thr Thr Ile Cys Ser Asn Ile Asn Leu Pro Ser Thr Leu
35 40 45
Ser Leu Ser Asn Ser Thr Ala Ala Pro Gln Ala Pro Gln Gln Gln Gln
50 55 60
Leu Gln Pro Ser Gln Cys Leu Val Arg Glu Gln Asp Gln Tyr Met Pro
65 70 75 80
Ile Ala Asn Val Ile Arg Ile Met Arg Arg Ile Leu Pro Pro His Ala
85 90 95
Lys Ile Ser Asp Asp Ala Lys Glu Thr Ile Gln Glu Cys Val Ser Glu
100 105 110
Tyr Ile Ser Phe Ile Thr Gly Glu Ala Asn Asp Arg Cys Gln Arg Glu
115 120 125
Gln Arg Lys Thr Ile Thr Ala Glu Asp Val Leu Trp Ala Met Gly Lys
130 135 140
Leu Gly Phe Asp Asp Tyr Val Glu Pro Leu Thr Leu Phe Leu Asn Arg
145 150 155 160
Tyr Arg Glu Met Glu Asn Glu Arg Ser Thr Ile Arg Asp Pro Ile Leu
165 170 175
Lys Arg Ser Ser Val Gly Val Val Asp Tyr Gly Asn Leu Gly Met Asn
180 185 190
Pro Phe Met Pro Thr Phe Pro Met Ile Pro Pro Pro Gln Gly Tyr Phe
195 200 205
Asp Ser Asn Met Leu Gly Gly Tyr Tyr Arg Asp Ala Pro Asp Gly Ala
210 215 220
Ser Gly Ala Ala Ser Gly Ser Asn Leu Ala Ala Ser Ser Ala Pro Asn
225 230 235 240
Ser Leu Leu His Phe Asp Pro Phe Ala Gln Phe Lys
245 250
<210> 153
<211> 198
<212> БЕЛОК
<213> Glycine max
<400> 153
Met Asn Met Asn Met Arg Gln Gln Gln Val Ala Ser Ser Asp Gln Asn
1 5 10 15
Cys Ser Asn His Ser Ala Ala Gly Glu Glu Asn Glu Cys Thr Val Arg
20 25 30
Glu Gln Asp Arg Phe Met Pro Ile Ala Asn Val Ile Arg Ile Met Arg
35 40 45
Lys Ile Leu Pro Pro His Ala Lys Ile Ser Asp Asp Ala Lys Glu Thr
50 55 60
Ile Gln Glu Cys Val Ser Glu Tyr Ile Ser Phe Ile Thr Gly Glu Ala
65 70 75 80
Asn Glu Arg Cys Gln Arg Glu Gln Arg Lys Thr Ile Thr Ala Glu Asp
85 90 95
Val Leu Trp Ala Met Ser Lys Leu Gly Phe Asp Asp Tyr Ile Glu Pro
100 105 110
Leu Thr Met Tyr Leu His Arg Tyr Arg Glu Leu Glu Gly Asp Arg Thr
115 120 125
Ser Met Arg Gly Glu Pro Leu Gly Lys Arg Thr Val Glu Tyr Ala Thr
130 135 140
Leu Ala Thr Ala Phe Val Pro Pro Pro Phe His His His Asn Gly Tyr
145 150 155 160
Phe Gly Ala Ala Met Pro Met Gly Thr Tyr Val Arg Glu Thr Pro Pro
165 170 175
Asn Ala Ala Ser Ser His His His His Gly Ile Ser Asn Ala His Glu
180 185 190
Pro Asn Ala Arg Ser Ile
195
<210> 154
<211> 240
<212> БЕЛОК
<213> Medicago truncatula
<400> 154
Met Glu Thr Gly Gly Gly Phe His Gly Tyr Arg Lys Leu Pro Thr Asn
1 5 10 15
Thr Asn Ser Ser Ala Val Ala Gly Thr Leu Lys Leu Ser Ser Val Ser
20 25 30
Glu Met Asn Thr Arg Gln Gln Val Gly Glu Gln Asn Asn Asn Gly Thr
35 40 45
Glu Gln Asp Asn Glu Cys Ile Val Arg Glu Gln Asp Arg Phe Met Pro
50 55 60
Ile Ala Asn Val Ile Arg Ile Met Arg Lys Ile Leu Pro Pro His Ala
65 70 75 80
Lys Ile Ser Asp Asp Ala Lys Glu Thr Ile Gln Glu Cys Val Ser Glu
85 90 95
Tyr Ile Ser Phe Ile Thr Gly Glu Ala Asn Glu Arg Cys Gln Arg Glu
100 105 110
Gln Arg Lys Thr Ile Thr Ala Glu Asp Val Leu Trp Ala Met Ser Lys
115 120 125
Leu Gly Phe Asp Asp Tyr Ile Glu Pro Leu Thr Met Tyr Leu His Arg
130 135 140
Tyr Arg Glu Leu Glu Gly Asp Arg Thr Ser Met Arg Val Glu Pro Leu
145 150 155 160
Gly Lys Arg Gly Met Glu Tyr Gly Asn Leu Gly Gly Phe Val Pro Gln
165 170 175
Phe His Ile Gly His Pro Asn Gly Gly Tyr Tyr Gly Asn Ala Ala Pro
180 185 190
Thr Tyr Met Met Arg Asp Gly Asn Asn Asn Asn Asn Asn Asn Asn Asn
195 200 205
Ala Pro Asn Ala Ala Asn Ala Ala Gly Gly Ser Ser His Ser Gln Ala
210 215 220
Leu Ala Asn Ala Glu Ala Asn Gly His His His His His Gln Tyr Lys
225 230 235 240
<210> 155
<211> 278
<212> БЕЛОК
<213> Zea mays
<400> 155
Met Asp Ser Ser Ser Phe Leu Pro Ala Ala Gly Ala Glu Asn Gly Ser
1 5 10 15
Ala Ala Gly Gly Ala Asn Asn Gly Gly Ala Ala Gln Gln His Ala Ala
20 25 30
Pro Ala Ile Arg Glu Gln Asp Arg Leu Met Pro Ile Ala Asn Val Ile
35 40 45
Arg Ile Met Arg Arg Val Leu Pro Ala His Ala Lys Ile Ser Asp Asp
50 55 60
Ala Lys Glu Thr Ile Gln Glu Cys Val Ser Glu Tyr Ile Ser Phe Ile
65 70 75 80
Thr Gly Glu Ala Asn Glu Arg Cys Gln Arg Glu Gln Arg Lys Thr Ile
85 90 95
Thr Ala Glu Asp Val Leu Trp Ala Met Ser Arg Leu Gly Phe Asp Asp
100 105 110
Tyr Val Glu Pro Leu Gly Ala Tyr Leu His Arg Tyr Arg Glu Phe Glu
115 120 125
Gly Asp Ala Arg Gly Val Gly Leu Val Pro Gly Ala Ala Pro Ser Arg
130 135 140
Gly Gly Asp His His Pro His Ser Met Ser Pro Ala Ala Met Leu Lys
145 150 155 160
Ser Arg Gly Pro Val Ser Gly Ala Ala Met Leu Pro His His His His
165 170 175
His His Asp Met Gln Met His Ala Ala Met Tyr Gly Gly Thr Ala Val
180 185 190
Pro Pro Pro Ala Gly Pro Pro His His Gly Gly Phe Leu Met Pro His
195 200 205
Pro Gln Gly Ser Ser His Tyr Leu Pro Tyr Ala Tyr Glu Pro Thr Tyr
210 215 220
Gly Gly Glu His Ala Met Ala Ala Tyr Tyr Gly Gly Ala Ala Tyr Ala
225 230 235 240
Pro Gly Asn Gly Gly Ser Gly Asp Gly Ser Gly Ser Gly Gly Gly Gly
245 250 255
Gly Ser Ala Ser His Thr Pro Gln Gly Ser Gly Gly Leu Glu His Pro
260 265 270
His Pro Phe Ala Tyr Lys
275
<210> 156
<211> 225
<212> БЕЛОК
<213> Arachis hypogaea
<400> 156
Met Glu Thr Gly Gly Gly Phe His Gly Tyr Arg Asn Leu Pro Thr Thr
1 5 10 15
Thr Ser Gly Leu Lys Leu Ser Val Ser Glu Met Asn Met Arg Ala Val
20 25 30
Glu Asn Asn Thr Gly Ser Ser Asn Asn Asn His Thr Asp Asp Asn Glu
35 40 45
Cys Thr Val Arg Glu Gln Asp Arg Phe Met Pro Ile Ala Asn Val Ile
50 55 60
Arg Ile Met Arg Lys Ile Leu Pro Pro His Ala Lys Ile Ser Asp Asp
65 70 75 80
Ala Lys Glu Thr Ile Gln Glu Cys Val Ser Glu Tyr Ile Ser Phe Ile
85 90 95
Thr Gly Glu Ala Asn Glu Arg Cys Gln Arg Glu Gln Arg Lys Thr Ile
100 105 110
Thr Ala Glu Asp Val Leu Trp Ala Met Ser Lys Leu Gly Phe Asp Asp
115 120 125
Tyr Ile Glu Pro Leu Thr Met Tyr Leu His Arg Tyr Arg Glu Leu Glu
130 135 140
Gly Asp Arg Thr Ser Met Arg Gly Glu Pro Leu Gly Lys Arg Thr Val
145 150 155 160
Asp Tyr Gly Thr Leu Gly Val Ala Ala Ala Ser Thr Phe Val Pro Pro
165 170 175
Phe His Ile Gly His His His His His Pro His Pro Ser Ser Tyr Tyr
180 185 190
Gly Thr Pro Met Gly Asn Tyr Ile Arg Asp Ala Pro Asn Ala Gly Ser
195 200 205
Ser Leu Gln Pro Pro Ser Leu Ala His Ala Glu Pro Asn Thr Gln Tyr
210 215 220
Lys
225
<210> 157
<211> 234
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 157
Met Glu Arg Gly Gly Phe His Gly Tyr Arg Lys Leu Ser Val Asn Asn
1 5 10 15
Thr Thr Pro Ser Pro Pro Gly Leu Ala Ala Asn Phe Leu Met Ala Glu
20 25 30
Gly Ser Met Arg Pro Pro Glu Phe Asn Gln Pro Asn Lys Thr Ser Asn
35 40 45
Gly Gly Glu Glu Glu Cys Thr Val Arg Glu Gln Asp Arg Phe Met Pro
50 55 60
Ile Ala Asn Val Ile Arg Ile Met Arg Arg Ile Leu Pro Ala His Ala
65 70 75 80
Lys Ile Ser Asp Asp Ser Lys Glu Thr Ile Gln Glu Cys Val Ser Glu
85 90 95
Tyr Ile Ser Phe Ile Thr Gly Glu Ala Asn Glu Arg Cys Gln Arg Glu
100 105 110
Gln Arg Lys Thr Ile Thr Ala Glu Asp Val Leu Trp Ala Met Ser Lys
115 120 125
Leu Gly Phe Asp Asp Tyr Ile Glu Pro Leu Thr Leu Tyr Leu His Arg
130 135 140
Tyr Arg Glu Leu Glu Gly Glu Arg Gly Val Ser Cys Ser Ala Gly Ser
145 150 155 160
Val Ser Met Thr Asn Gly Leu Val Val Lys Arg Pro Asn Gly Thr Met
165 170 175
Thr Glu Tyr Gly Ala Tyr Gly Pro Val Pro Gly Ile His Met Ala Gln
180 185 190
Tyr His Tyr Arg His Gln Asn Gly Phe Val Phe Ser Gly Asn Glu Pro
195 200 205
Asn Ser Lys Met Ser Gly Ser Ser Ser Gly Ala Ser Gly Ala Arg Val
210 215 220
Glu Val Phe Pro Thr Gln Gln His Lys Tyr
225 230
<210> 158
<211> 231
<212> БЕЛОК
<213> Brassica napus
<400> 158
Met Glu Arg Gly Gly Phe His Gly Tyr Arg Lys Phe Ser Leu Asn Thr
1 5 10 15
Thr Asn Pro Ser Glu Pro Ala Arg Phe Leu Met Ala Glu Gly Ser Met
20 25 30
Gln Leu Ala Glu Pro Asn Gln Thr Asn Lys Thr Ala Asn Gly Gly Glu
35 40 45
Glu Glu Cys Val Val Arg Glu Gln Asp Arg Phe Met Pro Ile Ala Asn
50 55 60
Val Ile Arg Ile Met Arg Arg Ile Leu Pro Ala His Ala Lys Ile Ser
65 70 75 80
Asp Asp Ser Lys Glu Thr Ile Gln Glu Cys Val Ser Glu Tyr Ile Ser
85 90 95
Phe Ile Thr Gly Glu Ala Asn Glu Arg Cys Gln Arg Glu Gln Arg Lys
100 105 110
Thr Ile Thr Ala Glu Asp Val Leu Trp Ala Met Ser Lys Leu Gly Phe
115 120 125
Asp Asp Tyr Ile Glu Pro Leu Thr Leu Tyr Leu His Arg Tyr Arg Glu
130 135 140
Leu Glu Gly Asp Arg Gly Val Gly Tyr Asn Ala Gly Ser Val Gly Met
145 150 155 160
Thr Ser Gly Met Val Val Lys Arg Pro Asn Gly Thr Met Gly Glu Tyr
165 170 175
Gly Ala Tyr Gly Val Val Pro Gly Met His Met Ala Pro Tyr His Tyr
180 185 190
Arg His Gln Asn Gly Tyr Ala Tyr Ser Gly Asn Glu Pro Asp Ser Lys
195 200 205
Met Gly Gly Pro Ser Ser Ala Ala Asn Gly Ser Arg Val Glu Leu Phe
210 215 220
Pro Thr Gln Gln His Lys Tyr
225 230
<210> 159
<211> 216
<212> БЕЛОК
<213> Phaseolus coccineus
<400> 159
Met Glu Ser Gly Gly Phe His Gly Tyr Arg Lys Leu Pro Asn Thr Thr
1 5 10 15
Ser Pro Gly Leu Lys Leu Ser Val Ser Asp Met Asn Asn Val Asn Thr
20 25 30
Ser Arg Gln Val Ala Gly Asp Asn Asn His Thr Ala Asp Glu Ser Asn
35 40 45
Glu Cys Thr Val Arg Glu Gln Asp Arg Phe Met Pro Ile Ala Asn Val
50 55 60
Ile Arg Ile Met Arg Lys Ile Leu Pro Pro His Ala Lys Ile Ser Gly
65 70 75 80
Asp Ala Lys Glu Thr Ile Gln Glu Cys Val Ser Glu Tyr Ile Ser Phe
85 90 95
Ile Thr Gly Glu Ala Asn Glu Arg Cys Gln Arg Glu Gln Arg Lys Thr
100 105 110
Ile Thr Ala Glu Asp Val Leu Trp Ala Met Ser Lys Leu Gly Phe Asp
115 120 125
Asp Tyr Met Glu Pro Leu Thr Met Tyr Leu His Arg Tyr Arg Glu Leu
130 135 140
Glu Gly Asp Arg Thr Ser Met Arg Gly Glu Ser Leu Gly Lys Arg Thr
145 150 155 160
Ile Glu Tyr Ala Pro Met Gly Val Gly Val Ala Thr Ala Phe Val Pro
165 170 175
Pro Gln Phe His Pro Asn Gly Tyr Tyr Gly Pro Ala Met Gly Ala Tyr
180 185 190
Val Ala Pro Pro Asn Ala Ala Ser Ser His His His Gly Met Pro Asn
195 200 205
Thr Glu Pro Asn Ala Arg Ser Met
210 215
<210> 160
<211> 312
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 160
Met Val Asp Glu Asn Val Glu Thr Lys Ala Ser Thr Leu Val Ala Ser
1 5 10 15
Val Asp His Gly Phe Gly Ser Gly Ser Gly His Asp His His Gly Leu
20 25 30
Ser Ala Ser Val Pro Leu Leu Gly Val Asn Trp Lys Lys Arg Arg Met
35 40 45
Pro Arg Gln Arg Arg Ser Ser Ser Ser Phe Asn Leu Leu Ser Phe Pro
50 55 60
Pro Pro Met Pro Pro Ile Ser His Val Pro Thr Pro Leu Pro Ala Arg
65 70 75 80
Lys Ile Asp Pro Arg Lys Leu Arg Phe Leu Phe Gln Lys Glu Leu Lys
85 90 95
Asn Ser Asp Val Ser Ser Leu Arg Arg Met Ile Leu Pro Lys Lys Ala
100 105 110
Ala Glu Ala His Leu Pro Ala Leu Glu Cys Lys Glu Gly Ile Pro Ile
115 120 125
Arg Met Glu Asp Leu Asp Gly Phe His Val Trp Thr Phe Lys Tyr Arg
130 135 140
Tyr Trp Pro Asn Asn Asn Ser Arg Met Tyr Val Leu Glu Asn Thr Gly
145 150 155 160
Asp Phe Val Asn Ala His Gly Leu Gln Leu Gly Asp Phe Ile Met Val
165 170 175
Tyr Gln Asp Leu Tyr Ser Asn Asn Tyr Val Ile Gln Ala Arg Lys Ala
180 185 190
Ser Glu Glu Glu Glu Val Asp Val Ile Asn Leu Glu Glu Asp Asp Val
195 200 205
Tyr Thr Asn Leu Thr Arg Ile Glu Asn Thr Val Val Asn Asp Leu Leu
210 215 220
Leu Gln Asp Phe Asn His His Asn Asn Asn Asn Asn Asn Asn Ser Asn
225 230 235 240
Ser Asn Ser Asn Lys Cys Ser Tyr Tyr Tyr Pro Val Ile Asp Asp Val
245 250 255
Thr Thr Asn Thr Glu Ser Phe Val Tyr Asp Thr Thr Ala Leu Thr Ser
260 265 270
Asn Asp Thr Pro Leu Asp Phe Leu Gly Gly His Thr Thr Thr Thr Asn
275 280 285
Asn Tyr Tyr Ser Lys Phe Gly Thr Phe Asp Gly Leu Gly Ser Val Glu
290 295 300
Asn Ile Ser Leu Asp Asp Phe Tyr
305 310
<210> 161
<211> 321
<212> БЕЛОК
<213> Brassica napus
<400> 161
Met Met Ala Asp Glu Asn Val Glu Thr Lys Ala Ser Thr Leu Ile Ala
1 5 10 15
Ser Val Gly His Gln Gly His Gly Phe Gly Ser Gly Ser Gly Gly His
20 25 30
His Gly Leu Ser Ala Ser Val Pro Leu Leu Gly Val Asn Ser Lys Lys
35 40 45
Arg Arg Met Pro Arg Gln Arg Arg Ser Ser Ser Ser Phe Asn Leu Leu
50 55 60
Ser Leu Pro Pro Pro Met Pro Leu Ser Pro His Val Pro Thr Pro Leu
65 70 75 80
Ser Ala Arg Lys Ile Asp Pro Arg Lys Leu Arg Phe Leu Phe Gln Lys
85 90 95
Glu Leu Lys Asn Ser Asp Val Ser Ser Leu Arg Arg Met Ile Leu Pro
100 105 110
Lys Lys Ala Ala Glu Ala His Leu Pro Ala Leu Glu Cys Lys Glu Gly
115 120 125
Ile Pro Ile Arg Met Glu Asp Leu Asp Gly Leu His Val Trp Thr Phe
130 135 140
Lys Tyr Arg Tyr Trp Pro Asn Asn Asn Ser Arg Met Tyr Val Leu Glu
145 150 155 160
Asn Thr Gly Asp Phe Val Asn Ala His Gly Leu Gln Leu Gly Asp Phe
165 170 175
Ile Met Val Tyr Leu Asp Leu Asp Ser Asn Asn Tyr Val Ile Gln Ala
180 185 190
Arg Lys Ala Ser Glu Glu Glu Glu Glu Glu Glu Asp Val Thr Ile Ile
195 200 205
Glu Glu Asp Asp Val Tyr Thr Asn Leu Thr Lys Ile Glu Asn Thr Val
210 215 220
Val Asn Asp Leu Leu Ile Gln Asp Phe Asn His His Asn Asp Asn Ser
225 230 235 240
Ser Asn Asn Asn Ser Asn Asn Asn Ile Asn Asn Asn Lys Cys Ser Tyr
245 250 255
Tyr Tyr Pro Val Ile Asp Asp Ile Thr Thr Asn Thr Ala Ser Phe Val
260 265 270
Tyr Asp Thr Thr Thr Leu Thr Ser Asn Asp Ser Pro Leu Asp Phe Leu
275 280 285
Gly Gly His Thr Thr Thr Thr Thr Asn Thr Tyr Tyr Ser Lys Phe Gly
290 295 300
Ser Phe Glu Gly Leu Gly Ser Val Glu Asn Ile Ser Leu Asp Asp Phe
305 310 315 320
Tyr
<210> 162
<211> 314
<212> БЕЛОК
<213> Medicago truncatula
<400> 162
Met Met Met Asp Glu Gly Glu Gly Lys Lys Lys Val Val Val Gln Lys
1 5 10 15
Thr Glu Ala Cys Gly Phe Met Ala Gly Val Glu Asp Glu Leu Gly Phe
20 25 30
Val Asn Val Lys Gly Asp Asn Asn Asn Gly Ser Gly Gln Arg Ile His
35 40 45
His Asp His Gly Phe Val Ala Ala Ala Phe Gly Thr Val His Arg Lys
50 55 60
Lys Arg Met Ala Arg Gln Arg Arg Ser Ser Ser Ser Thr Ile Thr Ile
65 70 75 80
His Leu Lys Asn Leu Pro Ser Ser Thr Thr Thr Thr Thr Thr Thr Thr
85 90 95
Thr Ser His Val Pro Ile Ser Pro Ile Pro Pro Leu Phe His Ser Leu
100 105 110
Pro Pro Ala Arg Glu Ile Asp His Arg Arg Leu Arg Phe Leu Phe Gln
115 120 125
Lys Glu Leu Lys Asn Ser Asp Val Ser Ser Leu Arg Arg Met Val Leu
130 135 140
Pro Lys Lys Ala Ala Glu Ala Phe Leu Pro Val Leu Glu Ser Lys Glu
145 150 155 160
Gly Ile Leu Leu Ser Met Asp Asp Leu Asp Gly Leu His Val Trp Ser
165 170 175
Phe Lys Tyr Arg Phe Trp Pro Asn Asn Asn Ser Arg Met Tyr Val Leu
180 185 190
Glu Asn Thr Gly Asp Phe Val Ser Thr His Gly Leu Arg Phe Gly Asp
195 200 205
Ser Ile Met Val Tyr Gln Asp Asn Gln Asn His Asn Tyr Val Ile Gln
210 215 220
Ala Lys Lys Ala Cys Asp Gln Asp Glu Tyr Met Glu Glu Ala Asn Asp
225 230 235 240
Thr Ile Asn His Ile Phe Val Asp Asp Tyr Glu Val Asn Lys Ser Cys
245 250 255
Phe Asp Val Ala Tyr Pro Ala Met Asn Asp Thr Ser Met Ser Phe Ile
260 265 270
Tyr Asp Thr Thr Ile Ser Asn Asp Ser Pro Leu Asp Phe Leu Gly Gly
275 280 285
Ser Met Thr Asn Tyr Ser Arg Ile Gly Ser Val Glu Thr Phe Gly Ser
290 295 300
Val Glu Asn Leu Ser Leu Asp Asp Phe Tyr
305 310
<210> 163
<211> 3275
<212> ДНК
<213> Arabidopsis thaliana
<400> 163
ggttggctat atggtccaaa ttttgatttg caatatgaga ttgcacagag agaacaatct
60
ttcattatga ttaattattg tacaagtaac aaacaccaat ctccgatata ctttggctct
120
ttagcacatt gttatgctag aagttagcgg aaatctatat gttgttaaac gcagcgttta
180
aattgaacag tgtaatttac cttgaaattt taagactaca tgctgtttag aatttcagat
240
gaaaacatct tgatgtttta gaaatccacg tgggaatagc gtaaaatctt atccaacgaa
300
cttattttgg ttttgttgta tttgtgcaag tcgtcacgct aatcgaaaaa agaaaagaaa
360
aaaagaagcc gtcatgatcg gccatttctc ggccgagtct gagtctgact ctgcgtccgt
420
gtcaccatta tcagatcgag cctgtcttat ctcgttgcga ttccctatgc aaaaatcttc
480
ttcttttttt tattccccca tttatctctg atctcttctc tcttctcaag taaacctctc
540
tgcttcacgt ctcttctttt cttgtcgatt ttccccagat aatcagttga aaacacaccc
600
aaattcatct tcgaatcaat aatggatata agtaatgagg ctagtgtcga tcccttttcg
660
attggaccat catctatcat gggtcgaacc attgctttca gagtcttgtt ctgtagatca
720
atgtcacagc ttaggcgtga tctctttcgg ttcttgttgc attggtttct tagatttaag
780
ctgaccgttt caccgtttgt gtcgtggttt catcctcgga accctcaagg gattttagcg
840
gtggttacaa tcattgcctt tgtgttgaaa cgatacacga atgtgaaaat aaaggcggaa
900
atggcttacc ggaggaagtt ttggaggaat atgatgcgga cggctttgac ttatgaggaa
960
tgggctcatg ctgctaagat gttagagaag gaaacaccaa agatgaatga atctgatctt
1020
tatgatgaag agttggttaa gaacaagctt caggagcttc gtcatcgtcg ccaagaaggc
1080
tcacttagag acattatgtt ttgtatgaga gctgatttgg tgaggaatct cggtaatatg
1140
tgtaattcgg agcttcataa aggtagactt caggttccta gacatatcaa agagtacatt
1200
gatgaggtgt ctactcagtt gagaatggtt tgtaactctg attcagagga gctttcttta
1260
gaagagaagc tttcttttat gcatgaaaca cggcatgcct ttggtagaac ggctttgctt
1320
ttgagtggtg gggcttctct tggtgcgttt catgttggtg tggttaggac tttggttgag
1380
cataagcttt tacctcgaat aattgctggt tctagtgttg gatccatcat ttgtgctgtt
1440
gtggcctcaa ggtcttggcc agaactacag agtttctttg agaattcttt gcattcttta
1500
cagttctttg atcagctcgg aggcgtgttc tcaatagtga aacgggtaat gacacaaggg
1560
gctctacacg atatcagaca gttgcaatgt atgcttagaa acctcacaag caatctcaca
1620
ttccaagaag cttatgacat gacaggaagg attctcggga tcaccgtttg ctccccaaga
1680
aagcatgaac ctcctcggtg tcttaactat ttgacttcgc ctcatgtggt tatatggagc
1740
gcagtgactg cttcttgtgc ttttcctggt ctctttgaag ctcaagagct aatggctaaa
1800
gatcgaagtg gagagatcgt accgtatcat ccacctttca atttggatcc agaagtaggc
1860
actaaatcat catctggacg ccggtggaga gatggtagtt tggaggttga tttaccaatg
1920
atgcagctta aagaactgtt caatgtcaat cattttattg tgagccaagc caatcctcac
1980
attgctccat tactgcgtct aaaggattta gttcgagctt atggtggtag attcgcagct
2040
aagctcgcgc atctagtgga gatggaggtc aaacatagat gcaaccaggt attagagctc
2100
ggttttcctc tcggtggact cgcaaagctt tttgctcagg agtgggaagg tgatgttaca
2160
gttgtaatgc ctgctactct tgctcagtac tcgaagatta tacaaaatcc gactcatgtc
2220
gagcttcaga aagcggctaa ccaaggaaga agatgcactt gggagaagct ctcagccata
2280
aaatcaaact gcgggatcga gcttgcgctt gatgattctg tagctattct taaccatatg
2340
cggaggctca agaaaagtgc ggagagagcc gccactgcca cgtcttcgtc tcatcacgga
2400
ttggcttcaa ccaccagatt caatgcttca agaagaatcc catcttggaa cgtccttgcc
2460
agagagaact caacaggctc actggatgat ctagtcactg acaataacct ccacgcttct
2520
tcgggcagga atttaagcga cagtgaaaca gagagcgtgg agttgagttc ttggacaaga
2580
actggtggac ctttaatgag aacagcttct gctaataagt tcattgattt tgttcagagt
2640
cttgatatcg acattgcatt ggtcagagga tttagtagca gtcccaattc tccagcagtt
2700
cctcctggtg gctcgtttac tccaagcccg agatccatag cggctcattc ggatatcgaa
2760
tcaaacagca atagcaacaa tcttggaaca agcacttcaa gcataacagt tactgaaggt
2820
gatcttctac agcctgagag aacgagtaac ggatttgtgt taaacgtcgt taaaagagag
2880
aacttgggaa tgccatcgat tgggaaccaa aatacagagt taccagagag tgtacagctc
2940
gatataccgg agaaggagat ggattgtagc tctgtatcag aacacgaaga agatgataac
3000
gacaatgaag aagaacataa cggctcgagt ctggttactg tttcttcaga agattccggt
3060
ttacaagaac cggtgtctgg tagtgttata gatgcttaga gtgtgattga ttcaagtgag
3120
tatagattct taattaaatt tgcagagttt ccaaagggtt tagtgcacca cttgtgtatg
3180
tttgtattgc ttattgtttg aaattcattt gtgaaatcga aatatatctg taaattcaga
3240
aaatattctc tcatccatta caaaatattt gagtc
3275
<210> 164
<211> 2795
<212> ДНК
<213> Brassica napus
<400> 164
tcccacgctc aggttctaat tgcaaaaaag gatcatactt tccttattaa aatcaatttc
60
ctgtgcttga tttctatctt aggaagctcg tagtagtttc tctgatagtg aatttgatga
120
aacaaagaaa aaaatgctga cttggtctca gattctaatt aaacacacac acacacataa
180
cctccaatgg atataagcaa cgaggccaat gtcgatccct tctcaatcgg accaacctcc
240
atcctcggcc gaaccatcgc cttcagagtc ctcttctgca aatcaatgct ccagctccgc
300
cgcgacctct tccgcttcct cctccactgg ttcctcacac tcaagctcgc cgtctccccc
360
tttgtctcct ggttccaccc ccggaacccc caggggatcc tcgccgtcgt cacgatcatc
420
gccttcgtcc tgaaacgcta caccaacgtg aaggccaagg ccgagatggc ctaccgtaga
480
aagttctgga ggaacatgat gcgcgcggcg ttgacttacg aggaatgggc tcacgccgct
540
aagatgttgg ataaagagac tccgaagatg aacgagtccg atctttacga tgaagagttg
600
gttaagaaca agctaatgga gcttcgtcat cgacgtcatg agggctctct tagagacatt
660
attttctgta tgagagctga tcttgtgaga aatctcggta atatgtgtaa ccctgagctt
720
cacaagggaa ggcttcacgt gccgagactc atcaaagagt atatcgatga ggtctctaca
780
cagcttagga tggtttgcga catggacact gaagagcttt ctctggagga gaaactttct
840
tttatgcatg agaccagaca cgcgtatgga agaacagctc tacttctcag tggaggagct
900
tctcttgggg ctttccatct tggtgtggtc aagacgcttg tggaacataa gctattgcca
960
agaattatag ctggttcaag cgtggggtct gtaatgtgtg cggttgtggg gacaaggtca
1020
tggcccgagt tgcagagctt ctttgaaggg tcctggcatg ctctgcagtt ctttgatcag
1080
atgggaggaa ttttcactac tgtgaagcgg gttatgactc aaggcgcagt ccatgagatc
1140
cggcatctgc aatggaagtt gaggaatctc accaacaatc tcacagtccg gaatttccgg
1200
gtcgacgact tcgaggatac tcgggataac ggtttgctca ccgacgaagc actagccgcc
1260
tcggtgctta actatctcac ttctcctcac gtggtgatat ggagcgcggt gactgcttct
1320
tgcgctttcc ctggtttgtt tgaagctcag gagctgatgg ctaaagatag gagtggggag
1380
atagtgccgt atcatccgcc ttttaatttg gaaccggagg aaggtgggga taagtcgtct
1440
acgaggaggt ggagagatgg gagtttggag gttgatttgc cgatgatgca gcttaaggag
1500
ctgtttaatg ttaatcattt tattgtgagc caggctaatc ctcacattgc tccgttgctg
1560
cgtttgaagg atatagttag agcttatgga ggtcgatttg cagcaaagct cgcgcaactc
1620
gcggagatgg aagtgaagca tagatgtaat caagtactag aactcgggct tcctctaaga
1680
gaagtagctt cactatttgc tcaagaatgg gaaggcgatg tcacaattgt catgccagct
1740
actttttctc agtacttgaa gatcatacaa gtcgacgatt tcgtcgagct tcaaaaagcc
1800
gctaaccaag gaaggagatg cacttgggag aagctatcag ccataaaagc aaactgtggg
1860
atcgagcttg cgcttgatga gtgtgtaact aatcttaacc atatgcgtag gctcaacaga
1920
agcgctgaga gagccgctgc tgctgctggc acgtcctcct cgtctcatca cggattagct
1980
tcaacgacaa gattcaatgc ttctagaaga atcccgtctt ggaacgtcat cgctagagag
2040
aactcaactg gctcactgga cgacctcgtc actgacagta acaataataa tctccacgcg
2100
gggaggaacc taagcgacag cgaaacggag agcgtggaga tgagttcttg gacgaggact
2160
ggtggaccgt tgatgagaac agcttctgct aataggttca ctgactttgt ccatggtctt
2220
gacgtggaca ttgcgttgac aagagggttt actagcagcc ctaactctcc agcggttcct
2280
ggcccggtta gtccgagttt tagtccaaga tcgagatcct tggcggctca atccgagagc
2340
gaatctgaca agagggaaag tagcaacagt tctagtatat cagctactga aggtgatctt
2400
ctgcagcctg agagaacgag taacggtttt gttttgaacg ttgttagaag agagaacttg
2460
gggatgcctg tggagaacca gagcggtgag ctgccggaga gtgtacagat agatatacct
2520
gagagggaga tggataatag ctctgtctca ggacatgaag atgataatga tgataatgat
2580
gatgaagaag aagaacataa gggctcggtt ccggttaaag attccggttt acaagattct
2640
tgtagtgtaa tagatgctta gactgatttg atccgagtga agagattctt gttcagcaaa
2700
gatcttggag tgttttagtg ctttgtaaat agtacaacta taggccgcaa gtaaggtgca
2760
tgttgtgtat gtttgcagtg attatgttga aaatt
2795
<210> 165
<211> 2670
<212> ДНК
<213> Brachypodium distachyon
<400> 165
atggaagaat ccggagaagc gagtattggg gccttcagga tcgggccgtc gacgcttctc
60
ggccgcggcg tcgcgcttcg cgtgctcctg ttcagctcgc tctggcgtct ccgggcgcgg
120
gcgcgcgccg ctgtgtcgcg cgtgcgcagg gccacgctgc caatggccgc gtcctggctt
180
cacctcagga acacccatgg cgtcctcctg attctcgtgc tcttcgggtt gctcctcagg
240
aagctctccg gtgcgcggtc gcggctggcg ctggcgcgcc ggcgtaggct gtgcaagagc
300
gcgatgcgct acgcggcgac gtatgagcag tgggtgcgtg ccgccaaggt gctcgacaga
360
atgtctgagc aggtgaacga gtctgatttt tacgacgagg agctgatcaa gagtaggctt
420
gaggagctcc ggaggcggag ggaggaaggg tcgctccggg atgtggtgtt ctgtatgcgc
480
ggcgatctcg tgaggaactt ggggaacatg tgtaatcctg agcttcataa gggcaggctc
540
gaggtgccca ggctgataaa agatttcatt gatgaggttt caactcagct gaaaatggtg
600
tgtgaatctg acaccgatgc gttatttttg gaagagaagc ttgcctttgt tcaggaaacc
660
aggcatgcct atgggaggac agcactactc ttaagtgggg gcgcttcact gggctctttc
720
catgtaggtg tagtgaaaac attagttgag cataagcttc tgcctcggat aatagcaggg
780
tctagcgttg gttccattat atgttcaatt gttgctactc gaacatggcc tgagattgag
840
agcttcttca tagactcatt acaaatctta cagttcttcg gtaggatagg tggaattttt
900
gctgtgacca aacgggttat gacttatggt gcacttcatg acattagcca gatgcaaagg
960
cttttgaggg atctcacaag taacttaaca tttcaagagg cttacgatat aactggccgt
1020
gttcttgggg tcactgtttg ctctcccaga aaaaacgagc cacctcgctg cctcaactac
1080
ctgacatcac cacatgttgt tatctggagt gctgtaactg cttcgtgtgc attccctggc
1140
ctctttgaag ctcaggaatt gatggcaaag gataggtttg gccacatagt tcccttccat
1200
gcgccctttt ccacagatcc agaacaaggt cctggagcat caaagcggcg atggagggac
1260
ggaagtttgg agatggattt accgatgatg caactaaagg agttattcaa tgtgaatcat
1320
ttcatcgtca gccaagctaa tcctcacatc tctccactcc tccgaatgaa ggagattgtc
1380
agatcctatg gaggtcgctt tgcgggaaag ctcgctcgtc ttgctgagat ggaggtgaag
1440
tatcgatgta accaagttct agaagttggc ctcccactgg gaggacttgc aaagttgttt
1500
gctcaggact gggagggtga tgtcactatg gttatgccag caacagtagc tcagtacttg
1560
aagattatac aagatccaac atatgcagaa ctccaaatgg ctgccaatca gggtcaaaga
1620
tgcacgtggg agaagctctc agcgatcaga gcaaactgtg caattgaact tgcattggat
1680
gaatccattg cggttctcaa ccacaaacga aggttaagaa gaagcacaag ggcagcagct
1740
tcttcccagg aatataccag caatgttcga ctcagaacac caaggagggt accctcatgg
1800
agctgcatca gtcgagagaa ttcgtcagga tctctctcag aagatcactt tgcggtcgct
1860
atttcatcca gtcaccaagg tactatacga gttgatggcg caccaaacat gcctcatcat
1920
gttcgtcaca gttcacacga tggaagtgag agcgaatcag aaaccattga cttaaattca
1980
tggaccagga gtggtgggcc tctaatgagg acttcatcag ctgatcagtt catcagtttt
2040
atccagaatc tcgagattga atctgagttc gatagggttc gtactacaga ggatgacaat
2100
acaggtattt tatcaggatc tacattttca aaagatccat acccaaacat tagttctaga
2160
gtcactacac cagatagatg cacagaagtt tctgaaacag agtcgtgcaa cgccggcaac
2220
acaagcatca ctgtttctga aggagatttg ctacaacctg agaggactac caccggaatt
2280
ctactcaatt ttgtcagaag agaagatctg cttggtcagc ataacagtga tgctgacatg
2340
accgaaagct ccttagccga agcatatgtg gacacatcac atttggaatc ttgtgatgcc
2400
atctcagcct ctgacagttc tgaaggtaac aaagacgcag ctgactcaga gaatctcttg
2460
gtttctcatg cagatttagt aacttcgcat caatcttcag ttgatgataa caaaggtggc
2520
tagattttga aagaattctt ttagtggctt gctaagtcga tgctgtacag gaaaaactgt
2580
agtgtctccg tttcgtgagc actactgctg gtagcatagt gaatattgta ctttgtacca
2640
gatactaaat aaatttgatt gcttgccatt
2670
<210> 166
<211> 3884
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Populus trichocarpa
<400> 166
gttttctttc ccttatccgc ctttgattgc aaaagtcaat gtcagagcca tcaccccctc
60
cttgctcaat ctttacgtaa ccgtatgtat atccttatct tcttctaaca tttcccaaga
120
ctccgatcct gtatttattg tattcacttc acccttcttc tctctttcct tcccgaacga
180
aaacaaagtc tcaatctttc attctctgtt tgtctaaagt ctgtacattc ttcactttct
240
cgagttgggt ttctttcttg aatttggttt cttgggtttg attttgtttt tcaagtggat
300
attgctattt attgggtggt gatattgaga cccttttgtt agttttgtat attggttttt
360
gaggtggatg tagttttttt aggggtttta gggtttggtt attgaaaact catatggcaa
420
ggttggcttc tggcaatctg gatttataag attctgtttt tcttgttgac acagtacagg
480
atcaaaaggg ttggattttt gttacttgtc aatatcttct tattttgtga tagctagtcc
540
ttttgcatta ggattgcata tctttattct atctacttca ttgtctctct atatattgcc
600
atcctatccg gggagagaca gattcaattg ttttattgtc cttctcattc tcattagaat
660
caaagtcttg acatacaatc ctttcacaat tgtgaaattt gattccttag tgaccatcta
720
ttgtagctgt ttcatatttg tttcgttcaa gctaattctg ttgttagatt tgagacaaaa
780
gaaggccccg cttccaatta cagaccactt tcttgttttg gttttagcta agatatggat
840
ataagcaatg aggccagtgt tgaccctttc aaaatcggac cttcatcgat cattggtagg
900
acaattgctt tcagagttct gttctgtaaa tcaatctcac atttgaggca aaaaatcttt
960
catgtgttgt tgaattacat ttatagagtt ggtgaatttg tggcgcctat gttatcatgg
1020
tttcatccaa ggaatccaca agggatattg gccatgatga cgataattgc atttttattg
1080
aaaagatatg cgaatgttaa attgagggcc gaaacagcgt ataggaggaa attttggagg
1140
aatacgatga gaactgcgtt gacatacgag gagtggtttc atgctgctaa aatgcttgat
1200
aaagagaccc caaagatgca tgaatgtgat ctctatgatg aagaactagt caggaacaag
1260
cttcaagagc tccaccaccg tcgccaagag ggatgtctta gagatataat cttttttatg
1320
agagccgatc ttgtaagaaa tctcggtaat atgtgtaacc ctgagcttca caagggtagg
1380
cttcaagtgc ccaagctcat aaaggaatat attgacgagg tctcaactca gttaagaatg
1440
gtttgtgact ccgattcaga ggagctttcg ttggaagaaa agcttgcttt catgcatgaa
1500
acgagacatg cttttgggag aacagctttg cttctgagtg gaggtgcttc acttggagcg
1560
tttcatgtgg gtgtggttaa aacactggtg gagcacaagc ttatgccccg aataattgct
1620
ggttctagtg tggggtcaat tatgtgttca gttgttgcca ccagatcgtg gccagagctg
1680
caaagttttt ttgaggattc ctggcactcg tttcagtttt ttgaccaatt gggtggaatt
1740
ttcacagttg tgaagagggt catgagacaa ggagctgttc atgaaatccg gcagttgcaa
1800
tggatgttaa ggcatcttac aagtaatctt acatttcaag aagcttatga catgactggt
1860
cgaattcttg ggatcacagt ttgctcacct aggaagcatg agccccctag atgccttaat
1920
taccttactt cccctcatgt tgttatatgg agtgcagtca ctgcttcttg tgcttttcct
1980
ggcctttttg aagcccagga actaatggca aaggacagaa gtggggaact tgtgccttat
2040
cacccaccct ttaatctgga tcctgaagaa ggatctgatg cacctatgcg taggtggagg
2100
gatggtagcc tggagattga tttaccaatg atacaattga aggaactatt caatgtcaat
2160
cattttattg taagtcaagc gaatcctcac attgctccat tgttgagact gaaggatata
2220
gtcagggcat atgggggtag ctttgctgcc aagcttgctc atctcgctga gatggaggta
2280
aaacatagat gcaatcaggt attggaactt ggttttccat taggtggact tgccaagctt
2340
tttgctcaag aatgggaagg tgatgttact gttgttatgc ctgccacact cgctcagtac
2400
tcaaaaatta ttcaaaaccc aaatcacttg gagcttcaaa aggcatcaaa ccaaggcaga
2460
aggtgcacat gggagaagct ttctgccata aaagctaatt gtggtattga gcttgctctt
2520
gatgagtgtg tttctgttct gaaccacatg cgtagactca aaaggagtgc tgagagagct
2580
gctgctgctt ctcatggcca agcaagctct gcgagcacat tgagatttag tgcttcaaaa
2640
agaattcctt cttggaattg catcgcaaga gaaaactcaa caggctcact tgaagaagac
2700
ttccttgctg atgttgcttc aacattccat caaggagttg gtgtggctgg aggaacttct
2760
actggtagga atttgagaac acaacgcaac ctacatcatg atggaagtga tagtgaatct
2820
gaaagtgtag atttgaattc ttggacaaga tctggcgggc ctttgatgag gactgcttct
2880
gcaaataagt tcattgactt tgtccaaagt ctggatgttg attctgagct aaggaaaggc
2940
ttcatggctc atcctaactc gcctggggct cagatgggag gcagggatcc atataatcag
3000
atctcaagag tgacaacccc agatagaaat tcagaaagtg agtttgatca gagagatttt
3060
agcaatagaa attctactgg tggttctagc attacagtca ccgaaggaga ttttttgcag
3120
cctgaaagaa tccataacgg gtttgtgctg aatattgtaa agaaagaaga tttggcacat
3180
cccaatagga tccatgattt ggagaattac aatagtgaag ttcctgaatg tgttcagctt
3240
gattgtcctg aaaaggacat ggatgctagc tcagaatcgg actatgctgc agaggaagac
3300
gactcccctg caacagattc cttgcataaa tcagcttcca ctcttgatca cacagatgat
3360
tctgtcgttc atgacattca ggagaagcat gtcgtggatg gttaactttg agtttcttct
3420
gcattactgt accaaaatat tgggtggagt tgattcccgg gttactgtca atcaaaggtt
3480
tccgactttc cgtcacaact ggagtatcat agacgagatt tagaatctgt ttatttttta
3540
ttttaaaaat atttttgaaa aaaattttga ttttattttg attttatttt tgttttaaat
3600
taatattttt ttggtgtttt tcatattatt ttgatatgtt gatattaaaa ataaattttt
3660
aatatcaatt attcaatcag atatattttt aagtaaaaca agacagtttg aaaagtaatc
3720
ggaactttta aaaggttgct cttagtagtg aattataaaa aacaattgaa agcaatctgg
3780
cagcgtcagg ctattgctgt tgtaaactaa ttttgtgcgc atactatgca acaattgtaa
3840
tccacatgct tagatttcag ccaacgagat ggaatttgac cctc
3884
<210> 167
<211> 2490
<212> ДНК
<213> Medicago truncatula
<400> 167
atggatcgta taagtaatga agccactgtt gatctttttc caatcggtcc ttcaggaatt
60
cttgcccgaa caattgcatt cagagtcctt ttctgcaaat ccatttcaca tttaaggtat
120
caattattct taactttatt cgattcgttt catagattta gaaaattctg gggacccatt
180
atatcatcct tgcatccaaa aaaccctcaa gggatattag ccatcatcac cattctcgct
240
ttcttgttaa aacgttacag taatgttaaa gtaagagctg aattagcata caggagaaaa
300
ttttggagaa atatgatgag atcagctttg acttatgagg agtgggctca tgcagctaag
360
atgcttgata aagagacgac attgaagacg atgaatgaat ccgattttta cgatgtagaa
420
ttggttagga ataaggttca agagttacga catcgtagac aagaggggtc tcttagagat
480
attatctttt gtatgagagc tgatcttgtt agaaatttag gtaatatgtg taaccctcag
540
cttcataaag gtaggcttca tgtgccgaga cagattaagg agtatattga tgaggtggcg
600
atgcagttga gaatggtttg tcattctgat tccgaggagc tttctttgga agaaaagctt
660
gctttcatgc atgaaactag acacgcgttt gggaggacgg ctttgttgtt gagtggtggt
720
gcttctcttg gagcttttca tgtcggtgta gttaaaacct tggtggaaca taaacttatg
780
ccgaggataa tttctggttc gagtgtagga tccattatgt gctctattgt tgctactagg
840
tcttggcctg agcttcaaag cttttttgaa gattcgttgc actcgttaca gttttttgat
900
caaatgggtg ggatttttac gattgtcaag agggttacaa catttggtgc agttcatgag
960
atcagacagt tgcagattat gttgaggcat ctaacgagca atcttacatt tcaagaagct
1020
tacgacatga caggtcgagt tcttgggatt acagtttgct ccccaaggaa gcatgaaccg
1080
cctagatgtc ttaactactt gacttcaccc catgttgtta tatggagtgc agtcacagct
1140
tcttgtgcct ttcctggtct ttttgaggct caggaattga tggcaaagga tagaagtgga
1200
gagattgttc cttaccatcc tccatttaat ttgggtcctg aagagggttc ctcacaagtg
1260
cggcgttgga gggatggtag cttggagatc gatctaccta tgatgcagtt gaaagaattg
1320
ttcaatgtca atcattttat tgttagtcag gccaatcctc atattgcgcc attattgaga
1380
ttaaaagaat ttgtacgagc ttatggaggt aattttgctg ccaagctggc tcatctggta
1440
gagatggagg ttaaacatcg atgtaatcaa atactggaac ttggttttcc attaggtgga
1500
cttgccaagc tgtttgctca ggactgggaa ggtgatgtga cagttgttat gcctgctact
1560
cttgctcagt actcaaaaat tatccagaac ccttcttatg tggagcttca gaaggcagct
1620
aaccaaggga gaagatgcac ttgggagaag ctttcagcca ttaaagcaaa ttgtggaatt
1680
gagcttgctc ttgatgagtg tgttgcaatt ctcaatcata tgagaagact caaaagaagt
1740
gccgagagag ctgcttctgc ttctcatggt ctttctagta ctgtcaaatt tagtgcttca
1800
aaaagaattc catcatggaa tgtcattgcg cgagagaatt ctacaggatc tcttgaagac
1860
tttcttgcag acactgctgc ttcatttcat cacggggtta gtagttccag tggagccacg
1920
ggtaaaaatt ccaagcacca ccgcagcatg catgatgtaa gtgacagtga atccgaaagt
1980
gctgaattga atacctggac cagatctggt ggtcctttga tgagaactgc ttcggcagat
2040
atgttcaccg actttgtcca aaacttagaa gttgatactg aactaaacag aggaatggga
2100
actaatttta gccctcgtga ttcccagtat cacagtccca gattaacaac accggataga
2160
tgctccgaga actcagaacc cgatcagaga gaaaatggca acaaggttgt catgaatgga
2220
tctagcataa tggtaactga aggtgatctt ttgcagcctg agagaatcca taatggaatt
2280
gtgtttaatg ttgtcaagaa agaagactta acaccttcaa gtaggagtca tgattatgat
2340
agtgaaattg ctgagtgtct ccaaattgaa tgtccaggga aggagatgga tgatgctgct
2400
agctcagctt cagaaaacgg agatgacgat tctgcaacag ctaggcccct aactgaaaca
2460
ccagactcta atcctacaga taattcctga
2490
<210> 168
<211> 2783
<212> ДНК
<213> Glycine max
<400> 168
atggatcata ttagtaatga ggccagtgtt gaccgttttc caattggtcc ttctggcatt
60
cttggtagga caattgcttt cagggttctt ttttgcaagt ctatctcaca ttttaggcac
120
cacatattta ttgtgttgtt agatctcttc tataggttta gggggggttt ggcatccttt
180
atatcatggt tgcatcccag gaaccctcaa gggatattgg caatgatgac aattgttgct
240
ttcttgttga aacgatacac aaatgtgaaa tcaagggctg aaatggcata taggaggaag
300
ttttggagaa acatgatgag aagtgctttg acctatgagg agtgggctca tgcagctaag
360
atgcttgata aagagacaac aaagatgaat gaatcagacc tttatgatgt ggaattggtg
420
aggaacaagc ttcaagagct ccgccaccgt cgacaagagg gatctctcgg agatataatg
480
ttttttatgc gtgccgatct tattagaaat ttaggtaata tgtgtaaccc tgaactacac
540
aagggtaggc ttcaggtgcc taaattaatc aaggagtaca ttgatgaagt aacgactcaa
600
ttgagaatgg tctgtgattc tgattcagag gagctatcat tggaagaaaa gcttgctttc
660
atgcatgaaa ctaggcatgc atttgggagg actgctttgc tgttaagtgg gggtgcctct
720
cttggagctt ctcatgtggg tgtagttaaa acactggtag aacataaact catgcctagg
780
ataattgctg gttcaagtgt gggatccatt atgtgtgctg ttgttgccac taggacttgg
840
cctgagctcc agagcttttt tgaggattca tggcactcat tgcaattttt tgatcaaatg
900
ggtgggattt ttgcagttgt taagagagtc acaacattgg gtgctgttca tgagatcaga
960
cagttgcaaa tgatgttgag gcatctaaca agcaacctta catttcaaga agcttatgac
1020
atgacaggca gaattcttgg gattactgtt tgttccccaa ggaagcatga accgcctaga
1080
tgtcttaact acttgacttc accccatgtg gttatatgga gtgcagtaac cgcttcttgt
1140
gcctttcctg gcctttttga ggctcaagaa ttgatggcaa aggatagaag tggagagatt
1200
gttccttacc accctccttt taacttaggc cctgaagagg gctccacacc agtgcgccgt
1260
tggagggatg gtagcttgga gattgattta cctatgatgc agttgaaaga actattcaat
1320
gtcaatcatt ttatagttag tcaggccaac cctcatattg caccactatt gagattgaaa
1380
gaatttgtgc ggacttatgg gggcaacttt gctgccaagc ttgctcatct tgtggagatg
1440
gaggtgaaac ataggtgtca tcaaatactg gaacttggtt ttccattagg tggacttgct
1500
aaattgtttg ctcaagactg ggaaggtgat gtgactgttg ttattcccgc aactcttgct
1560
cagtacacca aaattataca gaacccttct tatggagagc ttcaaaaggc agccaaccaa
1620
gggagaagat gtacctggga gaaactttca gccataaaag caaattgtgg cattgagctt
1680
gctcttgatg agtgtgttgt gattctcaat catatgagaa gactaaagag aattgctgag
1740
agagctgctt ctgcctctca tggtttgtcc agcactgtca ggttcagtgc ttcaaaaaga
1800
attccttcgt ggaattgcat tgcacgagag aattcgaccg gctcccttga ggaccttact
1860
gatgttgcct cctcattgca tcaaggcatc ggcagttcca gcagagccaa tggcaaaact
1920
tggaagaccc accgtggcat acatgatgga agtgacagtg actctgaaag tgttgatttg
1980
cattcttgga caagaactgg cgggcctttg atgagaacta cttcagcaaa tatgttcgtt
2040
gattttctcc aaaacttaga ggttgatacg gatcctaata aaggcttagt gagtcacact
2100
atccataatg attttcagta tcatagcccc aggctcacaa cactagatag gaactctgat
2160
agcacagaat ctgagccaag ggaaactggc aacagggttg tcaatgtgtc cagcatactt
2220
gtgaccgaag gtgatcttct gcagcctgaa aggatccata atgggattgt gtttaatgtt
2280
gtcaagaaag aagacttgtc acccttaagt agtagcagtc atggttttga aaattacaac
2340
attgaagttg ctgaatgtgt ccaagatgag tgtccaggga aggagataga tgctgctagc
2400
tctgcatctg aacacggaga tgatgaagaa tccatgccag ccaggtcctt aactgacatg
2460
ccagattaca attccattga tcatcattcg ggcacagatt cgggtatgga tcaaagcatt
2520
gttgacagtt agtgtcaagt atcagttctt ttccagtgac attttaatat tttgttccta
2580
ttgccctcca tattgtaaat agtactcatt ctagacttgg agaggtcttt attcatgatt
2640
ttgatgggaa tagcccacca atttggtttg ctcataaatg taacaaagat aaagagtttg
2700
tatacataaa ttccacgaca acattgatat ttcttggtta ccacttctca gatgaatgaa
2760
atggagacat ggttttcata att
2783
<210> 169
<211> 2724
<212> ДНК
<213> Sorghum bicolor
<400> 169
atggatgaca tcgccagcga ggcgccggtg ggggcgttcg ccatcggccc gtccacggcg
60
ctgggccgcg ccgtcgcgct ccgggtgctg ctctgcggct ccgcggcgcg cctgcggcac
120
cgcctggccg cggcgctccg cgccgcgctg cccgtcgcgg cggcgtggct gcacccgcgc
180
gacaacacgc gcgggatcct gctcgccgtc tgcgccgtcg cgctcctgct gcggggccga
240
cgcggcaggg ccgggctgcg ggcgagggtg cagtccgcct accgccgcaa gttctggcgg
300
aacatgatgc gcgccgcgct cacctacgag gagtgggcgc acgcggcgcg gatgctggag
360
cgcgaggccg ccccgcgccg cgccagcgac gccgacctct acgacgagga gctcgtccgc
420
aataagctcc gcgagctcag gcaccggcgc cacgagggat cgctcaggga catcgtcttc
480
tgcatgcgcg cggacctgct caggaacctc ggcaatatgt gcaaccccga actgcacaaa
540
gggaggctgc aggtgcctag actcataaag gaatacattg aggaagtatc tactcaactg
600
aaaatggtct gtgattctga ttcagatgag ttgcctcttg aagagaaact cgcatttatg
660
catgagacaa ggcatgcctt tggtagaaca gccttgctgc taagtggagg tgcttcattg
720
ggatcctttc atgtgggtgt tgttaaaacc ttggtagagc ataaactttt gccaaggata
780
atttcaggat caagtgttgg ctcgataatg tgttctatag tagcaacaag atcatggcct
840
gagctggaga gcttttttga agagtggcat tccctgaaat tttttgatca gatgggtgga
900
atctttcctg tggttaaaag aattttgacg caaggcgctg ttcatgatat aaggcacttg
960
caggtgcttt tgagaaacct taccagcaat ttgacatttc aagaagctta tgacatgact
1020
ggtcggattc ttgttgtcac cgtgtgttct ccaaggaagc atgagccgcc tcgatgccta
1080
aactatttaa catcacctca tgttcttatc tggagtgcag taacagcttc ctgtgctttt
1140
cctggacttt ttgaggccca agaattgatg gcaaaagata gatttggtca aaccattcct
1200
ttccatgctc cattcttatt aggcatagaa gaacgaactg ttgctccaac ccgccgctgg
1260
agagatggga gcttagaaag cgatttaccc atgaagcaat tgaaggaact attcaatgtg
1320
aatcatttca tagtaagcca agccaatcct cacatagctc cgctgttgag actaaaggaa
1380
atcgtcaggg cttatggagg cagcttcgct gccaagcttg ctgaacttgc tgagatggaa
1440
gtcaaacata ggtgtaatca agttttggaa cttggatttc ctctaggagg attagctaaa
1500
ttatttgctc aagattggga aggcgatgtt acagttgtta tgccagccac tcttgcgcag
1560
tattccaaga tgatacagaa cccatcttat gctgagcttc agaaggctgc gaatcaaggt
1620
aggagatgca cttgggaaaa gctatcagcc atcagggcaa attgtgctat tgagcttgca
1680
ctggatgaat gtgttgccct cctgaaccac ttgcgtaggc taaagaggag tgcagaaaga
1740
gcatccgcat cgcaaggata tggtccagca atcaggttct gcccatctag gaggattcca
1800
tcctggaatc tcatagcaag agaaaattca actggttctc ttgaagaaga aatgcttaca
1860
tctcctcaag gacctggagg agttgctgga acatctacca gaaaccagta tcctcagaga
1920
agtgcacatg agagcagcga cagtgaatct gagagtattg atttacactc ttggacaaga
1980
agtggtggcc ctcttatgag gacaacctca gccaataaat tcatcagctt tgttcagaat
2040
cttgagatcg acacagaatc cagaacaatt ccatcgaggg aagacataac tgatcttgtg
2100
acaccaaatg ctggtacctt ggcagctcat gcagtgagta gagaagcaat cgataggagc
2160
ttggacaatt cagctttaga tatccatgat accagtaccc ctagatcgac atttggccct
2220
tcaacaagta ttgtggtttc tgaaggtgac ttgttgcagc ctgaaaagat tgaaaatggt
2280
attttgttta atgttgtaag gagggatact ctgctcgggt ctagtagtgg agttgagtct
2340
caaggatctc ctcgggaacc agatgttgaa acagtacaga cggagtgcct tgatggcgtg
2400
tctacttctg atgatgatga tgacaaggaa ctaaatgcca ttgatgatgg aggaactagt
2460
cccatgagca gaaataatct acaacatcag gggtcctcac tggaagaaaa attataccat
2520
ccctcttcct taaattctga agacgagaca aacacaaaca aaccagaagc tgcatcgatt
2580
tttgatatat gtacagatat gcatccggca tctattagcc tacctgaagg gtcttcagaa
2640
aagacagaac tggaaacaac aaagattcct gatgacaatt cagctgttat gaatgatgaa
2700
gttgcctcag gtgctggtaa ctaa
2724
<210> 170
<211> 2985
<212> ДНК
<213> Zea mays
<400> 170
cattctctct ctctctctcc cttttcaatt tcgggggctt tatttctctc ctcccacgcc
60
ttgcatcttc ttgatgcgtt gcgtcccgac tcgaggcaga catcggaggc gcgccactga
120
cttagctcgc gatttctaga tccgcaaccc tgcgctgctc aactccgatt cttttagttc
180
ggattcgggt agcagcaggg cttcggtggc gatgacttga ttgatacgaa ttggggattt
240
cttgctgttt gcgcctctct tcgtatcggc gctgaagggc tcagctcgag attagaacaa
300
tttgggtttt ggggtctttt cttcttctac tactgctggt caagttcaca gaaatcgctg
360
gctccgtggt ccaatggacg agtccgggga agcgagcgtc ggctccttca ggatcgggcc
420
gtcgacgctg ctgggccgcg gggtggcgtt ccgcgtgctc ctcttcagct cgctgtggcg
480
cctgcgggcg cgcgcgtacg cggccatctc gcgcgtgcgc agcgcggcgc tgccggtggc
540
ggcgtcctgg ctgcacctca ggaacagcca cggcgtcctc ctcatggccg tgctcctcgc
600
cctcttcctg aggaaactct cggccgcgcg gtcgcgggcg gcgctcgcgc gccggcgcag
660
gcagcacgag aaggccatgc tgcatgccgg gacgtacgag gtctgggcgc gcgccgccaa
720
ggtgctcgac aagatgtctg agcaggtcca cgaggcggat ttctacgacg aggagctcat
780
caggaatagg ctcgaggaac tccggagacg gagggaggac gggtcgctcc gggacgtggt
840
gttctgtatg cgcggcgatc ttgttaggaa cttggggaac atgtgcaacc ctgaacttca
900
caagggcagg ctagaggttc ctaagcttat aaaggagtac attgaagagg tttctactca
960
actaagaatg gtgtgcgaat ctgacactga cgagttgctg ttggaagaga aacttgcctt
1020
tgttcaggag accaggcatg cctttgggag gacagcgcta ctcttaagtg ggggtgcttc
1080
actcgggtct ttccatgtag gtgtagtgaa aacattggtt gagcataagc ttctgcctcg
1140
gattatagca ggatcaagcg ttggttccat catatgttcg atcgttgcta cccgtacatg
1200
gcctgagatt gagagcttct tcacagactc attacagacc ttgcagtttt tcgacaggat
1260
gggcggaatt tttgcagtga tgaggcgtgt caccacttat ggtgcactgc atgacattag
1320
ccagatgcaa aggcttttga gggatctcac aagtaactta acatttcaag aggcttatga
1380
catgaccggc cgtgttcttg ggatcaccgt ttgctctcct agaaaaaatg agccaccccg
1440
ctgcctcaac tacctgacgg caccacatgt tgttatttgg agtgcagtaa ccgcctcttg
1500
tgcatttcct gggctctttg aagctcagga actgatggca aaggataggt tcggcaacat
1560
cgttcccttc catgcaccct ttgccacaga tcctgaacaa ggtcctggag catcgaagcg
1620
caggtggaga gacgggagct tggagatgga tttacccatg atgagactta aggagttgtt
1680
taatgtaaac catttcattg tgagccaaac taaccctcac atttctccac tcctccggat
1740
gaaagagctt gttagagcct atggagggcg ctttgctgga aagcttgctc gtcttgctga
1800
aatggaggtt aagtatcgat gcaaccaaat cctagagatc ggtcttccaa tgggaggact
1860
tgcaaaattg tttgcccagg attgggaggg tgatgtgacc atggttatgc cagcaacact
1920
tgctcagtac ttgaagatca ttcagaatcc aacatacgcg gagctccaaa tggctgccaa
1980
ccaaggccgc aggtgcacat gggagaagct ctccgcaatc agagcgaact gcgccattga
2040
acttgcactt gacgaatcca tagcggttct aaaccacaaa cggaggctaa aacgaagcat
2100
ggagaggacg gcggcggctt cgcagggtca ctctaactat gtccgaccca agactccgag
2160
gaggataccg tcatggagcc gcatcagtcg agagaactct ttggagtctc tctcggaaga
2220
gatctctgcg gttgctgctt cgtccatgca gcaaggcgct gctcttgttg tcggcgcacc
2280
accaacgact ctttctcagc atgttcggcg cagttctcat gacggaagtg agagtgagtc
2340
agaaaccatt gaccttaatt cctggaccag gagtggaggg cctctaatga ggacagcgtc
2400
cgccgacagg ttcatcagtt tcatccataa cattgagatt gacacagaat taagtaggcc
2460
ctgtgctgtg gaaggtgatg ctgcaggtat tttgtcagaa tctaccttcc caaacggtcc
2520
acgaccgaac aatagctcaa gtgttagtat gccaggtaga tgcacagaaa attctgggac
2580
cgagtcgtgc aacactgtca acaccagagc ttctactccc acaagcatgg ctgttcgtga
2640
aggagatttg ctgccgcctg aaagcactac tgataatgtc ctacttaaca ttgtgaaaag
2700
agacgccctg caggatggtg taactgaatt ggcggaaagc tcctgcgctg aaggatatgc
2760
ggcaaactgt gacaccgtct cagggctaga ctgctgaagg taacaagacg ctcgctgctg
2820
acttgagcaa tcaacaatta gctgatgatt agattcttct tgattttgat gatgaaaggt
2880
catttatatg tagctcacta cagcaacgca gtgtaggaaa attgtacctg ctcgatttaa
2940
actttaaaga gcatgccatg agtagctttg ttaatgttaa tattc
2985
<210> 171
<211> 1998
<212> ДНК
<213> Physcomitrella patens
<400> 171
atgaattact tagacactga cgccgacgct gcgctagagc atttcggcat tggacctctg
60
actttggcgc aaagagttgt ggcctttcgc gtcctatttt gtcgttgggt gaaagagctt
120
cgtgttgccc tcgcaaagag gctgcagcgg acacggaggg tatggagaca ggtgttctat
180
atgtggtttg ggtggttgaa ccctcgaaat cccagcgtcc ttctgttagc tgccgttgta
240
gcaaccatgc tcatgagaag agcgaaggca gggtctcaga aagcagagat tgcgtacaga
300
cggaagttct ggtccaattt aatgagggca gctttgacgt atgaggaatg ggctcatgcg
360
gcgcggatgc tagagaagga gcagaatcgg aggaaagatt cagacttgta cgatgaggat
420
ttggtgcgtt cgaagctcaa cgatcttcga ttgcgtcgtt tggagggtgg tgtggaggac
480
attcttttct gcattagggc cgatttagtg cgtaatttgg gtaacatgtg caatcccgaa
540
ctgcacaaag gccggctaca aactcccccc ctcatccagg aatacatcaa cgaagtgaga
600
taccatcttc gagctgtgtg tgggagcgac tcggacagct tcacacttga cgaaaaaatt
660
gcttttattc atgaaacccg ccatggtttt ggtcgcactg cacttcttct gagtggtgga
720
gcagctcttg gagcgtttca tcttggggtt gttcgaaccc ttgtcgagca tcgtttactt
780
ccccgagtga ttgccggtgc cagtgtggga tctgtcatat gctcatttgc tgcaactcga
840
acttggacag agctccagag ctttttcgaa gacaccatgc cccccatgca ctttttcgaa
900
aacatgggga gcatttttgc tattgcgcac aggcttctga ctcgaggtgc tgtgcatgaa
960
attggtatgc tgcaaaggaa aatgagacag ctcattgggg atttgacctt tcaggaagct
1020
tacgatctat ctggccgcgt gcttggaatc tctgtatgct cacctcggag actcgagcct
1080
ccgagatgtt taaattattt aacttctccc catgtagtca tttggagcgc agtcactgca
1140
tcctgcgcat tcccaggcct ttttgaagca caggagctga tggcgaagga tcgaactggt
1200
caacttgtac cctatcattc gccacctcag gttggccccg aggacaagga catggaaaag
1260
gggattggga agcggcgatg gcgagacggc agtctggaaa gcgatttgcc aatgatgcag
1320
ttgaaggaac tgtttaatgt gaatcatttc attgtcagcc aggcgaatcc gcatattaca
1380
ccatttttga ggttcaagga ttttgttcgt gcatatggag gagatttcgc tggaaaattg
1440
gcacacttag cggagatgga ggttaagcac cggtgcaagc agatgatgga gatgggcttt
1500
gaggtgtttg gattggctaa gctcttcgca caagattggg aaggagatgt cacgattgtg
1560
atgccggcca cttttgccca gtttgccaag atcatcacga acctgacagc cacagatctt
1620
cgcaaggcag tgatgcaagg ccgacgctgc acctgggcga agctatcagc cattcaggcc
1680
aactgtggca tagaattgat gctagacgaa tgtgtctctg aattaaaccg tcgtaggaaa
1740
gccctgcgtg aaatagagcg cagcgcaatg cagagcagcc atggtgggat gcgcgggtta
1800
tcaggaacaa agcgtatccc atcctggaac atcatcgccc gagagaattc ctgcggttcg
1860
ctagatgaag agagtcttca cgaggtgcgg atcccacatg atggtagcga cagcgacgat
1920
aatctggacc aaaatcagct ttcgtggacg agagcaggtg gcccgctcat gcggaccgca
1980
tcagcagcca aattcgtg
1998
<210> 172
<211> 3439
<212> ДНК
<213> Hordeum vulgare
<400> 172
gatcgcagtt agtttggctt gtacgtcgcg ttccccttcc acccttatct ccttctccgg
60
ctgaccggga cgccgcattt gtcccatcca cggcacggca cggcacgggc acgggaggga
120
gaagaagaag cccagctcga ctcctcctcc gcctcctcct ttcctctgat cccctccgtt
180
tgcccattcc ccagatccca gcacgccatg cccgggcgcg caggcgccaa gccgcaccgc
240
gcgcatttct cttccgccct gctccgatcc aaggccgcgg aggtgaccca gtgagctctc
300
ccgccacgcc cgtccgtccg ccggttcatc ggtcgcccat ggacgtcatc accaacgagg
360
cgcgcgtggg ggcgttcgcg atcggcccgt ccacggcggc gggccgggcg ctcgcgctgc
420
gcgtgctcct ctgcggctcc ctggcgcggc tgcggcaccg cctcgccgcc gcgctgcgcg
480
ccgcggcgcc cctggcggcg gcctggctgc acccgcgcca caacacgcgg gggatcctgc
540
tggccgtctg cgccgtcgcg ctcctgctgc gcggccgcgg gggccgcgcc ggggtgcgcg
600
cgcgcgtgca gtccgcctac cgccgcaagt tctggcgcaa catgatgcgc gccgcgctca
660
cctacgagga gtgggcgcac gccgcgcgga tgctcgagcg agagacgccg cgccgcgcca
720
ccgacgccga cctctacgac gaggagctcg tgcgcaacaa gctccgcgag ctcaggcacc
780
gtcgccagga gggctcgctc agggacatcg tcttctgcat gcgcgccgac ctgctcagga
840
accttggtaa catgtgcaac cccgagctcc acaagttgag gctgcaggtg cctaaactca
900
tcaaggaata cattgaggag gtatctactc aactgaaaat ggtttgcaat tctgattcag
960
acgagttacc tctcgaggag aaactggcat ttatgcatga gacaaggcat gcctttggta
1020
gatctgcctt actgctaagt ggaggagctt catttgggtc tttccatgta ggtgttgtga
1080
aaaccttggt agagcataag cttctaccta ggattatttc aggatcaagc gttggcgcaa
1140
taatgtgtgc tattgtcgcc acaaggtcat ggccagaact ggagagtttt tttgaggagt
1200
ggcattcctt gaaattcttt gaccaaatgg gtgggatctt tcctgtattt aaaagaattt
1260
tgacgcatgg ggctgttcat gacattaggc acttgcagac gcaattgaga aatcttacaa
1320
gcaacttaac atttcaagag gcatatgaca tgactggccg ggttctcgtt gttaccgtgt
1380
gttctccaag aaaacatgag ccacctcgat gcctgaacta tttgacgtca cctcacgttc
1440
tcatctggag tgcggtaact gcttcctgtg ctttccctgg actttttgag gcccaggagt
1500
tgatggccaa agatagattc ggagaaacag ttccttttca tgctccattc ttgttgggcg
1560
tggaggaacg agctgatgct gctacacggc gatggagaga tgggagctta gaaagtgatt
1620
tgcccatgaa gcagttgaag gaattattca acgtaaatca cttcatagta agccaagcca
1680
atcctcacat tgctccatta ctgagactaa aggagatcat cagggcttat ggaggcagct
1740
ttgctgcaaa gcttgctgaa cttgctgaga tggaagttaa gcataggttc aatcaagttc
1800
tggaacttgg atttccatta ggaggaatag ctaagttatt tgctcaacat tgggaaggtg
1860
atgtgacaat tgttatgcca gccactcttg ctcagtattc gaagatcata cagaatcctt
1920
cgtattctga gcttcagaaa gcagcaagtc agggtaggcg atgcacttgg gaaaagctct
1980
ctgccatcag ggcaaactgc gctattgagc ttgcattaga tgaatgtgtt gccctcctga
2040
accacatgcg taggctgaag agaagtgcag aaagagcagc cgcttcacaa ggatatggtg
2100
ctacaattag actctgtcca tctagaagga ttccgtcatg gaatctcata gcaagagaaa
2160
attcaactgg ttctctcgat gaggagatgc tcacatctcc cactgttaca agccatcaag
2220
cagttggagg gactgctggg ccatctaaca gaaatcacca tctccaacat agtatacatg
2280
atagcagtga cagtgaatct gagagtatag acttgaactc atggacgaga agtggtggcc
2340
ctctcatgag gacagcctcg gctaataaat tcatcagctt tgttcagaac cttgagattg
2400
acccagagtt cagaacaatt tcaccaaagg ggagtgaagg tgatattttg acaccgaata
2460
gtaacttgtt tgctggtcac ccaattggta gagagccagt tgataatcat ccaaggcctg
2520
ttactcctgg taggacctca ggcaatacag gttccgatcc tcatgatact cctgttccta
2580
ggtctccatt tggtctttcc gcgagtatca tggtccctga aggtgacttg ctgcagcctg
2640
aaaagattga gaatggtatt ttattcaatg ttgtccgaag ggatactctc ctagcgtcta
2700
ctagcggagt tgaacctcat ggatcttcac atgaggcaga tgtggaaact gtaccgaccg
2760
agtgccttta tggtgcttcg gatgacgacg acaacgtgga actgaatgcc aatgatgaag
2820
cgctatctga tcgtggagat cagagatctt cagttgcagg aaatctagat tcgtccgctt
2880
ccatggactg tcaagctgaa gcaagtacta ctcgatcaga agctccatct ctctttgata
2940
tctgtgtgga gattcctcca gcaaccatga ccacagaaaa tagtcggcct gacgagcctt
3000
cttcagacat aagactggag actgtaaaga cagaatgccc tgatgagaat tctgctgctg
3060
ggaatgctga agttgactca gttcctgcca gtaaagaatc ttcctattgg tctcagacat
3120
cagaaattgg acagcagcat caagtggata tgggatctgt gaactcctgt actgtttcat
3180
tttcagaaga tgatagacat gtgagcctta tttcgaacga gaaaccggtc actacttcca
3240
gtggcggagc tgagagtatg acatctggaa gaagtgaagc tgactagcat agaacttgcc
3300
tgttgaccga cctaatgttt ttctgtgttg ggacttggta gtttgaacaa ttcagcttga
3360
tctgatccat gctatgtgtg caatttaaac tcgtgtcacg atcaaactga attgtgtcta
3420
tatgtaggtg ttgtaatcc
3439
<210> 173
<211> 3470
<212> ДНК
<213> Nicotiana benthamiana
<400> 173
gttatctgat ccaaacttct gactttttct attttccgaa tccctatgtt ttttaataaa
60
tccatctctg ccattgcagt gatatattca tttattgtta tcaccttctt catttattgg
120
tccctctgtg ttttccatat attgaaggag aaaacattaa ctttatgcga ttttgtagtt
180
tttctggttg attcctacaa ccccttttga cattgatctt gtgggttaca aaaaacattg
240
aatctttatg tcaaaatttg atctttgtat ttcattttaa attgaaattt gatttttggg
300
ggtattaagg attcttttgt cggttgattt tgtgcctttt ttgccaagtt cttgtcggtc
360
tctgagctga atttccataa tttgacaaaa agaaaaggct aaagcagaaa ggttgggagt
420
ttctttcttt gactttcaga aactaaggta ttttctttga tctaattctt gttaatatct
480
ggttcaatct gattccgttg aatcttgtga atagcctttg tttccctatt gtcagaaaat
540
tatttccttt tcactttcct cgactctcag aagttagtac aatctttgtt ctgctaaatc
600
ttgtgaataa cctttagctt agagttttag gtatctgtat attgggttct cttaacattt
660
agcctagaag ccttctctag gattagtccc ccttttcatt gagatggata taagtaatga
720
ggctacaatt gacttctttt ccattggacc tactacgata ttgggtcgaa caatcgcctt
780
tagagtgttg ttctgtaaat caatttcaca attgaagcat cacctatttc atttcttgat
840
atattacttg tacaaattca agaatggttt gtcatactac ttgacaccct tgatctcgtg
900
gttgcaccct cgtaatccac aaggaatatt ggcattggta acgcttctcg ccttcttgtt
960
gaggcgatac acgaatgtaa aaatcaaggc tgagatggcc tataggagga agttttggag
1020
gaatatgatg agatctgcat tgacttatga ggagtgggct catgctgcca agatgctaga
1080
taaagagacc cctaaaatga atgaggcaga tctttatgat gtagaattag ttcgaaataa
1140
actccaagag cttcgacatc gtaggcaaga gggttctatg agggatatca tattctgtat
1200
gagagctgac cttgttagga atcttggtaa tatgtgtaat ccagaacttc acaagggaag
1260
gcttcatgtg cctagactga ttaaggatta tattgatgag gtttcaactc agttgagaat
1320
ggtatgcgac tctgattcgg aggagcttct cttggaagag aagcttgctt tcatgcatga
1380
aacaagacat gcctttggta ggacagcttt gcttttaagt ggaggtgctt ctttaggagc
1440
tttccatgtg ggcgtggtga aaacacttgt agaacacaaa ctgatgccac ggataattgc
1500
tggttcaagt gtcggctcga ttatgtgctc catagttgca actcgatctt ggcctgagct
1560
ccagagtttt ttcgaggact cctggcactc tttgcaattt ttcgatcagt tgggtgggat
1620
ttttactatt ttcaggaggg tcatgaccca gggtgctgta catgagatca gacagctgca
1680
ggtgctgtta cgtaatctca cgaataatct tactttccaa gaagcctatg acatgactgg
1740
tagagttctg gggattactg tttgctcgcc taggaaacat gaacctccta gatgcttgaa
1800
ctacttgact tcacctcatg ttgttatatg gagtgccgtt accgcttctt gtgcctttcc
1860
tggtctcttc gaagctcaag aacttatggc aaaggataga agtggagatc ttgttccata
1920
tcacccacca tttcatttgg gtcctgatgc cacttctagt gcatctgctc gtcgttggag
1980
ggatggtagc ttggaggttg atttgccaat gatgcagcta aaggagctct tcaatgtcaa
2040
tcactttatt gtgagccagg cgaatccgca tattgctcca ctgctgagga tcaaagagtt
2100
tgtaagagct tatggaggca actttgctgc caagcttgct caacttacgg aaatggaggt
2160
gaagcacaga tgcaatcagg tattagaact tggttttccc ttgggaggat tagcaaagct
2220
ttttgctcaa gaatgggagg gtgatgtaac tgttgtaatg cctgccactc tagctcagta
2280
ctcaaaaatc atacagaatc cctcgactct ggagctgcaa aaagcagcaa atcaaggaag
2340
aaggtgcact tgggaaaaac tctcagccat gaaagcaaac tgtggaattg agcttgcact
2400
tgatgaatgc gttgctatac tgaatcacat gcgtagactg aaaaggagtg ctgagagggc
2460
ggctgctgct tcacatggct tggcaagcac tgtcagattt aacacttcca gaagaattcc
2520
ttcttggaac tgcattgcac gagagaactc aacaggctcc cttgaagatt ttcttgcgga
2580
tgttgctgct tcacatcatc aaggaggcag tggttcgggg gcgcatgtta accgtagttg
2640
gcgaacgcac cggaatgcac atgatggtag tgacagtgag ccggaaaatg tggaccttaa
2700
ttcttggaca agatcgggtg gtcctttgat gaggacaaca tcagctgata agtttattga
2760
ctttgtccag aacttggaaa ttggttcgcg attgaacaaa ggattgacta ttgacctcaa
2820
caatattatt cctcagatgg caagcaggga ccatttctcc ccgagcccaa gggtaacaac
2880
acctgataga agttcagata cagaatttga tcaaagagat tttagttaca gggtccctgc
2940
gagtagttca agcattatgg taggcgaagg tgaccttctg cagcctgaaa ggactaacag
3000
cggtattgtc ttcaatgtgg taaggaaagg agacttgacc ccatcgaaca gaagccttga
3060
ttcagaaaat aatagttccg tgcaggatgc agttgctgag tgcgtgcaac ttgaaagtcc
3120
agaaaaggag atggatatta gctcagtatc ggaggatggt gagaatgatg ttgggcaagg
3180
aagtagggta aatgaagttg attgtagtaa aaatcgttca tcaatcggtg atggcaacga
3240
taagcaagtt attgatactt gagagtttag ctttgattat tctacacagg ccattcgaat
3300
tattttttat actcaaatgg agcttctttc agagctaaca cactcagaat tggggttgta
3360
aatagtgcaa gtagcaaatc tgtaataaat gtttagtgta gtcatcaccc ttctactagt
3420
tcaaagtggc tcagttcaat tcaaattcag aacttcgata attcatgttt
3470
<210> 174
<211> 713
<212> ДНК
<213> Nicotiana benthamiana
<400> 174
tgtatgagag ctgaccttgt taggaatctt ggtaatatgt gtaatccaga acttcacaag
60
ggaaggcttc atgtgcctag actgattaag gattatattg atgaggtttc aactcagttg
120
agaatggtat gcgactctga ttcggaggag cttctcttgg aagagaagct tgctttcatg
180
catgaaacaa gacatgcctt tggtaggaca gctttgcttt taagtggagg tgcttcttta
240
ggagctttcc atgtgggcgt ggtgaaaaca cttgtagaac acaaactgat gccacggata
300
attgctggtt caagtgtcgg ctcgattatg tgctccatag ttgcaactcg atcttggcct
360
gagctccaga gttttttcga ggactcctgg cactctttgc aatttttcga tcagttgggt
420
gggattttta ctattttcag gagggtcatg acccagggtg ctgtacatga gatcagacag
480
ctgcaggtgc tgttacgtaa tctcacgaat aatcttactt tccaagaagc ctatgacatg
540
actggtagag ttctggggat tactgtttgc tcgcctagga aacatgaacc tcctagatgc
600
ttgaactact tgacttcacc tcatgttgtt atatggagtg ccgttaccgc ttcttgtgcc
660
tttcctggtc tcttcgaagc tcaagaactt atggcaaagg atagaagtgg aga
713
<210> 175
<211> 1500
<212> ДНК
<213> Arabidopsis thaliana
<400> 175
cgaaaaaaga agtagaatat atatatatat atatatatat atatatatat atatatattc
60
gtgtggacat cataaatgcc taaatgataa tagttgattt cgagttttat tttcgttact
120
tccaatcaaa ttctccttgc accatattta tttttttact gtgagaacat atataagtat
180
atattggaat tacgtatccg agaggttttt gcatatttcg tttatttatt ttcgatatcc
240
acactactgt attattaaaa atttgaaaaa ttcaactagg gcttttcatc ttctctagaa
300
ttattcgttt atttatgtcg atgtccacac tattattaaa ataaaacgag aggatatggt
360
tggatcatcc aagtttcgtt tatgactctt tgttcattta caaacgttta gttttccact
420
taagttttga aaagagttaa tttccaatat attcggcaca gtttttcaag tgtattcatc
480
tgtttttttt ttttttggtt ggctatatgg tccaaatttt gatttgcaat atgagattgc
540
acagagagaa caatctttca ttatgattaa ttattgtaca agtaacaaac accaatctcc
600
gatatacttt ggctctttag cacattgtta tgctagaagt tagcggaaat ctatatgttg
660
ttaaacgcag cgtttaaatt gaacagtgta atttaccttg aaattttaag actacatgct
720
gtttagaatt tcagatgaaa acatcttgat gttttagaaa tccacgtggg aatagcgtaa
780
aatcttatcc aacgaactta ttttggtttt gttgtatttg tgcaagtcgt cacgctaatc
840
gaaaaaagaa aagaaaaaaa gaagccgtca tgatcggcca tttctcggcc gagtctgagt
900
ctgactctgc gtccgtgtca ccattatcag atcgagcctg tcttatctcg ttgcgattcc
960
ctatgcaaaa atcttcttct tttttttatt cccccattta tctctgatct cttctctctt
1020
ctcaagtaaa cctctctgct tcacgtctct tcttttcttg tcgattttcc ccagataatc
1080
aggtaaataa ggctactttc ttatttgatc tggtggtctt tgtgttgaaa tctctgggtt
1140
ttctctgttg atttcaaagt tctctctttt tttttttgtt tactgggtgc tgtgaaaaat
1200
gatcttgtca aagtctcctc ttttcatcga attgaaactc taattagaaa aaagatcata
1260
acttttatta aaaaaatgag tttgctttgc ttaattttgc gaattgcttc atagattcat
1320
tgattagcct atttggggta acaaaaaaaa gctgacacgg tttcagattc caaaaataga
1380
tcatgactct gtttcttctc tgcagaggtt ttaataaata tatgcttctt ctcatgagtt
1440
ctcgtttttt ttgtcacctt cgcagttgaa aacacaccca aattcatctt cgaatcaata
1500
<210> 176
<211> 2871
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность, комплементарная гену
pSSU-Олеозина в Т-ДНК pJP3502. В порядке (комплементарной
последовательности): терминатор лектина Glycine max 348 нт, 3?-экзон
255 нт, интрон UBQ10 304 нт, 5?-экзон 213 нт, промотор SSU
<400> 176
ggcccctaga atctaattat tctattcaga ctaaattagt ataagtattt ttttaatcaa
60
taaataataa ttaataattt attagtagga gtgattgaat ttataatata ttttttttaa
120
tcatttaaag aatcttatat ctttaaattg acaagagttt taaatgggga gagtgttatc
180
atatcacaag taggattaat gtgttatagt ttcacatgca ttacgataag ttgtgaaaga
240
taacattatt atatataaca atgacaatca ctagcgatcg agtagtgaga gtcgtcttat
300
tacactttct tccttcgatc tgtcacatgg cggcggcccg cggccgcttc attactcgag
360
ccaggaggat ggatcgatgc tggtctgaga ccctgctacc ggttgctgac tgaactgctc
420
ggcacggtcc ttcatttcac gggccttgct cgccaacttt gtcttggccg actccaactg
480
atccgctccg ggtggatgtt tccccgtcag gtaacggtag atccaggaca gcacagacag
540
agcggcaaca ccaaatcccc cgcttgccag aaaacccgct cccaacagga agatggtgat
600
gactgcagat cagaaaaact cagattaatc gacaaattcg atcgcacaaa ctagaaacta
660
acaccagatc tagatagaaa tcacaaatcg aagagtaatt attcgacaaa actcaaatta
720
tttgaacaaa tcggatgata tctatgaaac cctaatcgag aattaagatg atatctaacg
780
atcaaaccca gaaaatcgtc ttcgatctaa gattaacaga atctaaacca aagaacatat
840
acgaaattgg gatcgaacga aaacaaaatc gaagattttg agagaataag gaacacagaa
900
atttacctgc agggaccagt acaggcgaga agatcaccag gagaggtgtg gcgattgtca
960
gcgcaatgac cgttccagcc agggtcaacc cggataacac caacaggcta cctccggcag
1020
taaccgcggt cgctgccttt acaacacgct gagcacgcgg ttgcagttgc aagtgggggg
1080
cacgtgtttg ttgctgctgc ccgtagtgct ctgccatggt tttttttaac ggagcaagcg
1140
gccgctgttc ttctttactc tttgtgtgac tgaggtttgg tctagtgctt tggtcatcta
1200
tatataatga taacaacaat gagaacaagc tttggagtga tcggagggtc taggatacat
1260
gagattcaag tggactagga tctacaccgt tggattttga gtgtggatat gtgtgaggtt
1320
aattttactt ggtaacggcc acaaaggcct aaggagaggt gttgagaccc ttatcggctt
1380
gaaccgctgg aataatgcca cgtggaagat aattccatga atcttatcgt tatctatgag
1440
tgaaattgtg tgatggtgga gtggtgcttg ctcattttac ttgcctggtg gacttggccc
1500
tttccttatg gggaatttat attttactta ctatagagct ttcatacctt ttttttacct
1560
tggatttagt taatatataa tggtatgatt catgaataaa aatgggaaat ttttgaattt
1620
gtactgctaa atgcataaga ttaggtgaaa ctgtggaata tatatttttt tcatttaaaa
1680
gcaaaatttg ccttttacta gaattataaa tatagaaaaa tatataacat tcaaataaaa
1740
atgaaaataa gaactttcaa aaaacagaac tatgtttaat gtgtaaagat tagtcgcaca
1800
tcaagtcatc tgttacaata tgttacaaca agtcataagc ccaacaaagt tagcacgtct
1860
aaataaacta aagagtccac gaaaatatta caaatcataa gcccaacaaa gttattgatc
1920
aaaaaaaaaa aacgcccaac aaagctaaac aaagtccaaa aaaaacttct caagtctcca
1980
tcttccttta tgaacattga aaactataca caaaacaagt cagataaatc tctttctggg
2040
cctgtcttcc caacctccta catcacttcc ctatcggatt gaatgtttta cttgtacctt
2100
ttccgttgca atgatattga tagtatgttt gtgaaaacta atagggttaa caatcgaagt
2160
catggaatat ggatttggtc caagattttc cgagagcttt ctagtagaaa gcccatcacc
2220
agaaatttac tagtaaaata aatcaccaat taggtttctt attatgtgcc aaattcaata
2280
taattataga ggatatttca aatgaaaacg tatgaatgtt attagtaaat ggtcaggtaa
2340
gacattaaaa aaatcctacg tcagatattc aactttaaaa attcgatcag tgtggaattg
2400
tacaaaaatt tgggatctac tatatatata taatgcttta caacacttgg attttttttt
2460
ggaggctgga atttttaatc tacatatttg ttttggccat gcaccaactc attgtttagt
2520
gtaatacttt gattttgtca aatatatgtg ttcgtgtata tttgtataag aatttctttg
2580
accatataca cacacacata tatatatata tatatatatt atatatcatg cacttttaat
2640
tgaaaaaata atatatatat atatagtgca ttttttctaa caaccatata tgttgcgatt
2700
gatctgcaaa aatactgcta gagtaatgaa aaatataatc tattgctgaa attatctcag
2760
atgttaagat tttcttaaag taaattcttt caaattttag ctaaaagtct tgtaataact
2820
aaagaataat acacaatctc gaccacggaa aaaaaacaca taataaattt g
2871
<210> 177
<211> 1578
<212> ДНК
<213> Arabidopsis thaliana
<400> 177
gtcacacaca cataaacact ccacacgctc tgcttcgtcc aatcaccaaa cacgctttaa
60
tgactctcac gttttcctcc tccgccgcaa ccgttgccgt tgctgctgca accgtaacct
120
cctccgctag ggttccggtt tatccactcg cttcgtcgac tcttcgtgga ttagtatctt
180
tcagattaac cgcgaagaag ctgtttctgc cgcctcttcg ttctcgcggc ggcgttagtg
240
tgagagccat gtctgagctt gttcaggata aagaatcgtc cgtcgcggcg agcattgctt
300
tcaatgaagc cgccggtgag acgccgagtg agcttagtca ttcccgtact ttcttggatg
360
cgcgaagtga acaagatctt ttatctggta tcaagaagga agctgaagct ggaaggttgc
420
cagcaaatgt tgcagcagga atggaagaat tgtattggaa ctacaaaaat gcagttttaa
480
gtagtggagc ttccagggca gatgaaactg ttgtatcaaa catgtctgtt gcttttgatc
540
gcatgcttct tggtgtggag gatccttata cttttaatcc atatcataaa gcagtcagag
600
aaccatttga ctactacatg tttgtccata catacatccg tcctcttatt gatttcaaaa
660
attcgtacgt tggaaatgct tctatattct ctgagctgga agacaagatt cgacagggac
720
acaatatcgt gttgatatca aaccatcaaa gtgaagctga tccggctgtc atttctctat
780
tgcttgaagc acaatctcct ttcataggag agaacattaa atgtgtggct ggtgatcgag
840
tcatcactga tcctctttgt aagccgttca gtatgggaag gaacctcata tgtgtttact
900
cgaaaaagca catgaatgat gatcctgagc ttgttgacat gaaaagaaaa gcaaacacac
960
gaagcttaaa ggagatggct acaatgctaa ggtctggcgg tcaacttata tggattgcac
1020
caagcggtgg aagggaccgc ccgaatcctt ctactgggga atggtttcct gcaccctttg
1080
atgcttcttc ggtagacaac atgagaagac tggttgaaca ttctggcgct cctggacata
1140
tatatccaat gtctttgctt tgctatgaca tcatgccccc tccaccccag gttgagaaag
1200
aaatcggaga gaaaagatta gttgggtttc acggtactgg actatcaatt gctcctgaaa
1260
tcaacttctc agacgtcaca gcagactgcg agagccctaa tgaggcgaaa gaagcataca
1320
gccaagcttt gtacaagtcg gtgaatgaac aatacgagat cttaaactct gcgattaaac
1380
acagaagagg agtagaagca tcaacttcaa gggtctcttt gtcacaacct tggaattagt
1440
ctctcgtttt agggtaacac tttcaaaact cataaatctt ctgtctcaga agttttgttg
1500
caactgtata tatattgaga gagagagcat tgttctttca tttgcaggat acacaaacac
1560
aatcaatgga aaatactc
1578
<210> 178
<211> 459
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 178
Met Thr Leu Thr Phe Ser Ser Ser Ala Ala Thr Val Ala Val Ala Ala
1 5 10 15
Ala Thr Val Thr Ser Ser Ala Arg Val Pro Val Tyr Pro Leu Ala Ser
20 25 30
Ser Thr Leu Arg Gly Leu Val Ser Phe Arg Leu Thr Ala Lys Lys Leu
35 40 45
Phe Leu Pro Pro Leu Arg Ser Arg Gly Gly Val Ser Val Arg Ala Met
50 55 60
Ser Glu Leu Val Gln Asp Lys Glu Ser Ser Val Ala Ala Ser Ile Ala
65 70 75 80
Phe Asn Glu Ala Ala Gly Glu Thr Pro Ser Glu Leu Ser His Ser Arg
85 90 95
Thr Phe Leu Asp Ala Arg Ser Glu Gln Asp Leu Leu Ser Gly Ile Lys
100 105 110
Lys Glu Ala Glu Ala Gly Arg Leu Pro Ala Asn Val Ala Ala Gly Met
115 120 125
Glu Glu Leu Tyr Trp Asn Tyr Lys Asn Ala Val Leu Ser Ser Gly Ala
130 135 140
Ser Arg Ala Asp Glu Thr Val Val Ser Asn Met Ser Val Ala Phe Asp
145 150 155 160
Arg Met Leu Leu Gly Val Glu Asp Pro Tyr Thr Phe Asn Pro Tyr His
165 170 175
Lys Ala Val Arg Glu Pro Phe Asp Tyr Tyr Met Phe Val His Thr Tyr
180 185 190
Ile Arg Pro Leu Ile Asp Phe Lys Asn Ser Tyr Val Gly Asn Ala Ser
195 200 205
Ile Phe Ser Glu Leu Glu Asp Lys Ile Arg Gln Gly His Asn Ile Val
210 215 220
Leu Ile Ser Asn His Gln Ser Glu Ala Asp Pro Ala Val Ile Ser Leu
225 230 235 240
Leu Leu Glu Ala Gln Ser Pro Phe Ile Gly Glu Asn Ile Lys Cys Val
245 250 255
Ala Gly Asp Arg Val Ile Thr Asp Pro Leu Cys Lys Pro Phe Ser Met
260 265 270
Gly Arg Asn Leu Ile Cys Val Tyr Ser Lys Lys His Met Asn Asp Asp
275 280 285
Pro Glu Leu Val Asp Met Lys Arg Lys Ala Asn Thr Arg Ser Leu Lys
290 295 300
Glu Met Ala Thr Met Leu Arg Ser Gly Gly Gln Leu Ile Trp Ile Ala
305 310 315 320
Pro Ser Gly Gly Arg Asp Arg Pro Asn Pro Ser Thr Gly Glu Trp Phe
325 330 335
Pro Ala Pro Phe Asp Ala Ser Ser Val Asp Asn Met Arg Arg Leu Val
340 345 350
Glu His Ser Gly Ala Pro Gly His Ile Tyr Pro Met Ser Leu Leu Cys
355 360 365
Tyr Asp Ile Met Pro Pro Pro Pro Gln Val Glu Lys Glu Ile Gly Glu
370 375 380
Lys Arg Leu Val Gly Phe His Gly Thr Gly Leu Ser Ile Ala Pro Glu
385 390 395 400
Ile Asn Phe Ser Asp Val Thr Ala Asp Cys Glu Ser Pro Asn Glu Ala
405 410 415
Lys Glu Ala Tyr Ser Gln Ala Leu Tyr Lys Ser Val Asn Glu Gln Tyr
420 425 430
Glu Ile Leu Asn Ser Ala Ile Lys His Arg Arg Gly Val Glu Ala Ser
435 440 445
Thr Ser Arg Val Ser Leu Ser Gln Pro Trp Asn
450 455
<210> 179
<211> 2455
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Populus trichocarpa
<400> 179
agtgcgggtg attgggtgag gagtgaagac gctgatttta gaggttttga gagagtggca
60
gtctgcagag aataggaatc cgaccatatc ctccaaaacc cgcgctggac tcagtcaccg
120
ccaatcatca atcagccacc catcaacacc aaaaatcccc gtccttttga tttccaccac
180
ataaaaatag cacactgctc ctccttcact ccattcctat cttaataata ataataataa
240
taaagctcaa ctcttctctt ctaagtcaag acatgatcct ttccattcct gctccttcgt
300
cggcattctt cacaactact aaaccgtctc caccttttcc tagggtttct aaactctgct
360
tcttaacccc ctcatattct ctttcccttc gttttagatc cactgctcga cgctccactt
420
cttttccttg tgtcctctct tctctcaacc ttcacgcaat ggctgaactc gttcaggata
480
aagaagtctt cgcttctgct gaagttgatt acagcaagaa gaaaaacagg actcgttctc
540
gctcgtttct tgatgcaaca actgaacaag agttactgtc gggaatcagg aaggaatcag
600
aagcaggaaa acttccttca aatgttgctg caggaatgaa agatctgtat cagaactaca
660
aaaccgcagt tttgcaaagt ggaattccca acgcacatga gattgtattg gaaaatatgg
720
ctgctgcatt ggatcttata ttctttgatg ttgaggaccc gtttatcttc tcaccttatc
780
acaaagcttt gagaaagcca tatgactact ttgaatttgg tcaaaagtat atccgtccat
840
tgattgattt tagaaattca tatgtaggca atgtttccat tttcaatgaa attcaagaga
900
agcttcggca gggtcacaat attgtcttga tatcaaacca ccaaactgaa gcagatccag
960
ctgtcattgc actgttgctt gaaacatcaa gccctcacat tgctgaaaac ttgatctatg
1020
ttgctgggga tagagttgtc acagatcctc tttgcaagcc attcagcatg ggaaggaatc
1080
ttatatgtgt atactcaaaa aagcacatga atgatgaccc tgaacattca gaggagaaga
1140
gaaaagcaaa tatccgaagt ttgaaagaga tggctttgct tttaaggggt ggctcacaaa
1200
tagtctggat tgcaccaagt ggtggcaggg accgtccaga tcccttgtca ggagagtggt
1260
atccggcaca ctttgatgct tcttcagtag acaacatgag aaggcttgct gaacattctg
1320
gagctccagg acatgtttat cctctggcac tattatgcca tgacatcatg ccccctccgc
1380
ctcaggtgga aaaggaaatt ggagagagaa gagttatttc atttcatgga gttggattat
1440
cagttgcacc agaaatcagc ttctctgaag ttacagcggc atatgaaaat cctgaagagg
1500
ctaaggaggt atatacagag gctctgtata agtctgtgac tgagcaatac aatgtgctta
1560
aatctgctgt acatggaaaa caagggctag gggcgtccat tccaactgtt tctttgtcac
1620
agccatggaa ttagtcaacc ttttctatac ttgattaggc caatagtttt gttatatagt
1680
tctgcaactc ctggaccaca attctagcgg tccttctagt caagtatgtg ccaggagaag
1740
cttctctctc catgatgata tggatggctt tttctggaga tgcaatctaa gctacaagtt
1800
tttgctgtgc ttacattcta tcaaagccaa atctcacaca atatcttgaa gccaaattca
1860
tctgaaacgc gagctgttcc agaggttcaa tttcaggtgt gcagataaca gttcctagta
1920
aacacaagag ctagtcgtct gaggcgatat acatgtatat tttctcaatt ttttggtggc
1980
cgatcatatt ctttttacac caattgctca attgctactc atttttctcc ctcgttcacc
2040
ttcaataact agaagttttc atgctataac acttgcacac agaagtacta tgaacagagt
2100
tggagcacat tttgcctctt gactaaacaa gacttgtttt tagctgccac accaaacttt
2160
ttatatgatg caattatggt agtcgttttc tcttgttttg gtcaaaaccc aaaccagcta
2220
tagttgctac agccaatcga gagtggtgca tgtttgtttg tttttttttt tttttttgtc
2280
ctcagttata gtaaccatgt tcaactgaac tatgcatctc ttaggacacc acctcttaag
2340
ccccgtgatc taaccgtgtt ttcgaatttt tttttttttt ttgggctttt ggtttattta
2400
aacgcagcag ctttgaccca agttaaaaca aaaaaatcta ttaaaaaaat tgtag
2455
<210> 180
<211> 1389
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Jatropha curcas
<400> 180
atgacacttt ctgcttttcc ttccacattc ctctttagaa tacaatcgcc atcaacgcct
60
agggtttcca tttccctccc ttccttatct tcaaagctct gtttggttcc tccctctttt
120
tctcctcctt cgcttgctct taaatcgagt gcgcgaagga ccatttgtcc ttgcttgctc
180
tcttctctca acgccaacgt ggctcacctt ctcaaggagg aaaaagaagt tgtggcttcg
240
gcttccggct gcgagaagga ggaggaaaag aagatggaac agcctagtca ctcccgcact
300
ttcctgcatg ccagaacgga acaagatttg ctgtctggaa ttagaaaaga agcagaagca
360
gggaggttgc cttcaaatgt tgcagcaggg atggaagaat tgtatcagaa ttatagaaat
420
gcagtgatac aaagtggaac ccccaatgca gaagagatca tactgtcaaa tatggccgtt
480
gctttggatc gtataagctt ggatgttgag gacccttttg tcttctcaca ttatcacaga
540
gcattgagag agccgtttga ctactataac ttcggtcaaa attatattcg tcctttggtt
600
gattttagaa attcttatgt tggcaatatt tcccttttcc atgaagtgga agagaagctt
660
cagcagggtc ataatattgt cttgatgtca aatcaccaaa ctgaagcaga cccggctata
720
attgcattgc tgcttgagaa aacaaagccc tatattgctg agaatttgat ctatatagca
780
ggtggtagag tcataacaga tcctctttgc aagccattca gcatgggaag gaatcttata
840
tgcgtgtact caaaaaaaca catgaatgat gttcctgagc ttactgagat gaagaaaaga
900
gcaaacatac ggagtttgaa ggagatggcc attccattaa ggggtgggtc acgaatagtg
960
tggattgccc caagtggtgg tagggaccgc ccagatcatc tgactggaga atggtatcca
1020
gcaccatttg atgcttcttc agtggataac atgagaaggc ttgctgaaca ttctggtgct
1080
cctgggcata tttatccatt ggcattatta tgccatgaca taatgccccc tccccttcag
1140
gtgcaaaagg aaattggaga gaaacgagtg atctcctttc atggggttgg attatcaatt
1200
gcaccgggaa tcagcttctc tgaaattgcg ggtagttgtg aaaatcctga agaggcaaag
1260
aacatttatt cacaacttct gtatgattca gtgactgcgc aatacaacgt gcttaaatct
1320
gccataaatg gcaaacgagg gctagaggct tcaattccaa ctgtctcttt gtcacaacca
1380
tggaattaa
1389
<210> 181
<211> 1368
<212> ДНК
<213> Ricinus communis
<400> 181
atgattcttt ccattctttc ccctacacta ccatcgccta gggtttgtat ttccatttct
60
tctgtatctt caaagctctc tctagtccct gtctcttctt tttctcttcc tcctcctttg
120
gccatagtaa gatggtcatc aaggtcctcc atttgtcctt gtttcttctc ttcttctctc
180
aacgccaatc cagtccccga actcctcaac gatgataaga agaagaacaa caacaacaac
240
aagagcaaga agggaaagtg tactcctcac tcccgcactt ttcttgatgc aagaactgaa
300
caagagttgc tgtatggaat taggaaggaa gcagatgcag ggaggttgcc tttaaacatt
360
gcagcaggga tggaagaagt ttatcggaat tatagaaatg cagttttgca aagtggaatt
420
ccaaatgcaa aagaaatcat actgtcaaat atggctgttg cgttagatcg tatgtgcttg
480
gatgttgagg acccttttgt cttctcacct tatcataaag cactaagaga accattcgat
540
tactataatt ttggtcaaaa ttatatccgt cctctgattg attttaggaa ttcatatgtt
600
ggcaacattt cgcttttcca tgaagttgag cagaagcttc agcagggtca caatattatt
660
ttgatgtcaa accaccagac tgaagcagat ccagctgtca ttgcattgtt gcttgaaaaa
720
acaaatccct acattgctga gaatttgatc tacgttgcag gtgatagagt tgtaacagat
780
actctatgca agccattcag catgggaagg aatcttatat gtgtgtactc gaaaaaacac
840
atggctgatg ttcctgagct tactgagatg aagaaaaaag caaacattcg cagtttaaag
900
gagatggtca tgattttaag ggatgggtct caaattgttt ggattgctcc aagtggtggc
960
agggaccgcc cagattcttt gactggagaa tggtgtccag caccctttga tgcttcttca
1020
gtggataaca tgagaaggat tactgaacat tctggcgctc caggacatat ttttccatta
1080
gcgttgttat gccacgatat catgccccct ccacctgagg tacaaaagga aattggagaa
1140
agaagaatga tctcctttca tggagctgga ttatctattg cacccgaaat cagcttctct
1200
gaaattgctg ttgcttgcga agatcatgaa gaggctaaga acgcatatgc acaggtttta
1260
tatgattctg tgactgagca atacaatgtg cttaaatctg ccatacatgg aaaacaagga
1320
ctagaggcat caacttctac cgtctcattg tcgcaaccat gggattag
1368
<210> 182
<211> 1344
<212> ДНК
<213> Helianthus annuus
<400> 182
atgtcgattc tcccgtcttc ttctcctact ctcttcttct ccaccgcaaa ccctagggtt
60
tctgtttctc tttcacttac ttctacagtt tctacatctt catccgtgcg cagtcgctcg
120
attttccggc attttccgta cctagcgttt tctagggcag cgaatgccgc cgcggagacg
180
tttgaaggca agaagtggtc gtcgtcctcc gctacacaac cgatctccgg atccgagctc
240
ggttactcgc atacattcat cgatgctctg tctgaacaag atcttctttc tgtaattcaa
300
agagaggtag aagctggagc actgccaaaa catatcgctc actcaatgga ggaactctat
360
cagaactaca aaaatgcggt tttccaaagt ggtaatccct gtgcagaaga tactgtattg
420
tcaaacatgc gtgtagcatt tgatcgaatg ttcttggatg tgaaggagcc tttcgaattt
480
tcaccgtatc atgaagctat tcgagagcct tttaattact atatgtttgg tcaaaattat
540
attcgtcctc tgatcaattt cagggaatca tatgttggca acgtctctct tttcagtgaa
600
atggaagaac aactgaagca gggtgaaaat gtaattttga tctcaaacca ccaatccgaa
660
gcagatccag ctgtcattgc cttgttgctt gaaacaacaa atccttatat ttccgagaac
720
ataatctatg tggcagggga cagagttata acggatcctc tttgtaagcc tttcagcatg
780
ggaaggaact tgctgtgcgt atattcaaaa aaacatatga acgatgttcc tgagcttgct
840
gatatgaaaa ggagagcaaa tacaagaagt ttaaaagaga tggctttgct tttgaggggt
900
ggatcaaaaa taatatggat tgcaccaagt ggtggaaggg acaggcctga tcccgtcaca
960
aatcaatggt ttccagcacc attcgatgcc agttctctgg acaacatgag aaggcttgtg
1020
gaccatgctg gtgtggtggg tcatatatat cctttagcca tactatgcca tgacatcatg
1080
ccccctcctc ctcaggttga gaaagaaatt ggagagaaaa ggttgatatc ttttcatggc
1140
actggaatat cagttgcacc tgaagttgat ttccaaaacg ccactgcttc ttgtggatcc
1200
cccgaggagg ccaaggcagt ttattcacag gcactttatg attcagtgtg cgagcaatac
1260
aacgtgctac aatccgccat aaatggagca aaaggcttag aagcatcaac atcaagtgtc
1320
tcattgtcgc aacctgttga ctag
1344
<210> 183
<211> 1374
<212> ДНК
<213> Medicago truncatula
<400> 183
atgtttacaa caccattttc ttctccttca accgcatttt tctctccacc taaagcctca
60
tattcttctt cttcttcttc ttcttcttct tcttcttcgt tacctcttcg tagttctttc
120
actttttatc atcttcgatt taatgcaaca acttcttctt cttctgtaac aacttctgga
180
acttcttctt cttcatattg ttctcctctt gctttcaatt ctaataataa aaaacctaaa
240
gaaatttctg ctaatatggc ggcttcttct gtttcttctc gcactttcct caatgccaga
300
aatgaacaag atgttctttc tggaattaag aaggaagtag aagccggaac tttgcccccc
360
actattgctg aagggatgga agaattgtac cttaactata aaagtgcagt tgttaaaagt
420
ggagatccca aagcagatga gattgtattg tcaaatatga ctgctttatt agatcgcata
480
tttttggatg tgaaggagcc ttttgtcttt gaagcacacc ataaagcaaa gagagagcct
540
tttgactact acatgtttgg ccaaaattat attcgtccct tagttgattt caacacttct
600
tacgttggca acatgcccct tttcatacaa atggaagagc aacttaagca gggacacaat
660
attatcttga tgtcaaacca ccaaagtgaa gctgatccag ctattattgc attgctgctt
720
gaaatgcgac ttccacatat tgctgaaaac ttgatttatg tggcaggaga tagagttata
780
accgatcctc tatgcaagcc cttcagtatt ggcaggaatc tgatctgtgt ttattcaaaa
840
aagcacatgc ttgatgatcc agcacttgta gagacgaaaa gaaaagcaaa tacacgaagt
900
ctgaaggaaa tggccacgct tttaaggagt ggatcacaaa taatttggat tgccccaagc
960
ggtggtaggg atcgaccagt tgccaactct ggggaatggg caccggcacc ctttgattct
1020
tcttcagtgg acaatatgcg aaggcttgtc gatcattcag gtccaccagg tcatatctat
1080
cctatggcaa tactgtgcca tgacataatg ccccctccac taaaggttga aaaagaaatt
1140
ggggagaaaa gaattatatc atatcatggg actggcatat cacttgctcc agaaataagc
1200
ttttccgaca tcactgcttc ttgtgaaaat cctgaaaagg ctaaagaagc atactcgaaa
1260
gccttgtatg attctgtgac tagtcaatat gatgtgctgg agtctgccat acacggcaaa
1320
aaaggattag aagcatcaac tcccgcagtt tccttgtcgc agccatggaa gtag
1374
<210> 184
<211> 1967
<212> ДНК
<213> Glycine max
<400> 184
ggctgagact gaggagcgga tcctatctct ctttcacaca ctctccttct ctttcgtatg
60
aagaatgagc acgaccggtt cttcggctta ccactgtgtg gcacacctcc caaataataa
120
gactatgttt atgctctcta cgccgccaac aaccacattc ttcgctacgc ctagggttct
180
tccgtttctc tcttcaaaac tttcttcttc ttcttcttct tctactgcgt cgtcctcgcc
240
ttgttgctcc tccatcactc ccaaggttaa atccaaagat aacaacaatt gctacctcgt
300
ctccgctaaa cattctcccg ctaacatgtc cgcttcggtt tcgtcacgca ccttcctcaa
360
cgctcggaac gaacaagagc ttctagctgg aatcaggaaa gaagtagaag ctggatctct
420
gcctgctaat gttgctgcag gaatggaaga agtgtacaat aactataaaa gtgcagttat
480
ccaaagtgga gatcccaagt caaaggagat tgtattgtcg aatatgattg ctttattgga
540
tcgcatattc ttggatgtga cggatccttt tgtctttcaa ccacaccaca aagcaaagag
600
agagcctttt gactactacg tgtttggtca gaattatatc cgtcctttag ttgatttcaa
660
aaattcttat gttggcaaca tgcccctttt cattgaaatg gaagagaaac ttaagcaggg
720
acacaacatc atcttgatgt caaatcacca aactgaagct gatccagcca tcattgcttt
780
gctgctcgaa acacgactcc catatattgc tgaaaacatg acctatgtag caggagatag
840
agttataact gatcctctgt ccaaaccatt cagtattggc aggaatctca tttgtgttta
900
ctctaaaaag cacatgcttg atgatccagc tcttgtagag atgaaaagaa atgcaaatat
960
acgagctctg aaggaaatgg ctatgctttt aaggagtgga tcacaaatag tctggattgc
1020
cccaagtggt ggaagggatc gcccagatcc ccacaccgga gaatgggcac cggcaccctt
1080
tgatacttct tcggtagata atatgagaag acttgttgaa cattctggtc caccgggcca
1140
tgtatatcct ttggcgatat tgtgccatga tataatgccc cctccactaa aggttgagaa
1200
agaaattggg gagaaaagaa ttatatcctt tcatgggact ggcatatcag tggctccagc
1260
attaagcttt tctgaaacta ctgctactag tgaaaatcct gaaaaggcta aggaggtatt
1320
cacaaaagcc ctgtatgatt ctgtgacgga gcaatataat gtgctgaaat ctgcaataca
1380
tggcaaaaaa ggatttgaag catcaactcc agtagtttct ttgtcacagt catggaagta
1440
gatgaaatct gcatttcttc attgcaattt gctctgatgc agaagcaagt tacaagactt
1500
cagtcaaaca atttcaactg attcacttct gagggactgc ctattactac accggtcacc
1560
gaatgattta gcttgttgga agtttgcagt caaatacata tttttcattt catttttcct
1620
tttgctcttg gttgccgtta tcagcattca attcatctgg aatctgtttc agttcagaag
1680
gttcaaattc tgctgcttac tgtacaggtc tctcttagtt cggtgtcaga tttggttcgt
1740
tgactgataa aatactaaat tttttaccta caattttgtg atcaggctta gctagctgaa
1800
tagataaaat ataattggtt ccatttgtat tttaagtcaa ctttgttcca ttatagatga
1860
atagatgtta gtattacatg ttcagacggg gtcagtgaat aaactggtcc aaatgctaat
1920
gcaaaattat tcatattggt aaaataaaag ctctacagtt accgtta
1967
<210> 185
<211> 1674
<212> ДНК
<213> Carthamus tinctorius
<400> 185
tctctctctc tcacacacaa cacacaaaac acacactact gctactttct ctctctacta
60
cactctcctc tcgctatgtc gatcttcttc tctccttcct cccctactct cttcttctcc
120
accacaaacg caaatcctag ggtttctcct tcatcttcac cttcttctgc cttcactcct
180
cctctgtctt cttctcgcct ccgcccgatt ctccgggggt ttccgtgcct cgcgttctct
240
gcgccggcga atgccgccca tggcacggcg gagaccgtcc acggcaataa gtggccgtca
300
ccgtcgtcct cctcctctgc tgctacgcaa ccgtccgctg gatccgacca cggtcactct
360
cgtacattca tcgatgctcg ttccgaacaa gatcttcttt ctggaattca aagagagttg
420
gaagctggaa cactgccaaa acatattgct caagcaatgg aggagctata tcagaactac
480
aaaaatgcag ttctccaaag tgcggctcct catgcagaag atattgtgtt gtcaaacatg
540
cgtgtagcgt ttgatcgtat gttcttggat gtgaaggagc cgtttgaatt ttcaccatat
600
catgaagcta ttttggaacc ttttaactac tatatgtttg gtcaaaatta tattcggcct
660
ttggtcaatt tcagggaatc atacgttggc aatgtctccg ttttcggtgt aatggaagag
720
cagcttaagc agggtgacaa ggtggttttg atctcaaacc atcaaacaga agcagatcca
780
gctgttattg ccttgatgct tgaaacaaca aacccccata tttctgagaa cataatctac
840
gtggcagggg atagagtaat aacagatcct ctttgcaagc ctttcagcat gggaaggaat
900
ctgttgtgcg tgtattcaaa aaagcatatg aatgatgttc ctgagcttgc tgagatgaaa
960
aaaagatcaa atacaagaag tttaaaaggg aggatggctt tgcttttgag gggcggatct
1020
aaaataatat ggattgcgcc aagtggtggc agggacaggc cagatcctat cacaaatcag
1080
tggtttccgg caccgtttga tgccacttcg cttgacaaca tgagaaggct cgtggaccat
1140
gctggtttgg tgggtcacat atatccttta gccatattgt gccatgacat catgccccct
1200
cctcttcagg ttgagaaaga aattggagag aagagttgga tctcttttca tggcaccgga
1260
atatcagtgg caccggaaat taatttccaa gaagttactg cctcttgtgg gtcccccgag
1320
gaggcgaagg cagcttattc acaggcactc tatgattccg tgtgtgaaca atacaaggtg
1380
ctacattctg cggtacatgg aggaaaaggg ttagaagcat caacaccaag tgtctcgttg
1440
tcacaaccct tgcagtttct cgattagtct cttggtttag aggaggtgaa agcatattct
1500
tttgtttaga tgacataggt gtatagatga taccgaagaa tagatgtaca aacaagtgat
1560
agaaagatgt atgtctaatc aaaaaatgtt ttctgcatct tgtaaaggga tcttcaaaac
1620
agacctttta ttttagctgc agcaaccaat atatcaaaac aggtttttct tttt
1674
<210> 186
<211> 1893
<212> ДНК
<213> Solanum tuberosum
<400> 186
cgcacatatt catttcactc actttctttc ccgacccttt ctctctctaa agctctccag
60
tctgtggtga tgttgatcct ctcagcggct tcgtcttctt cttcctcctt catgctttct
120
tccgcttcgt cttcttctgc acgcattccg aggcagttat cttcattttc aacttgtgtt
180
ccagtagtag taacaactgt ttcttctgca gcaacttcga ctctatttcc gatttcctgc
240
ttcggtgtga aatcgaggac tgttgggatt cggaagctgc ggtgtgccgt tttttgtgct
300
tcgaaggtac gtggaatggc agaaatgatt gaagatgcca tgacggtttc tgcttctgag
360
agccatgagc ttccgcagtc ccgagacttc cttgacgcac gcactggaga agacttgcta
420
tctgctgttc aaaaagctgt ggaagatgaa aaactgccgc ttaatgttgc tgaaggaatg
480
gaggatttgt atcagaacta tcggaatgca gttttacaaa gtggagtccc caaagcagat
540
gaggccactt tgtataacat ggctcttgta tttgatcgtg tttttgtgga tgtgaaggat
600
ccttttgaat tctcgccata tcataaggcc attcgtgaac cttttgacta ttacaagttt
660
ggtcaaaatt atatccgcca gctagttgat ttcaggagtt cttatgttgg gaatatctca
720
gttttcggtg aaatggcaga gaagcttaaa cagggtgata atgttgtctt gatgtcaaac
780
catcaaagtg aagccgatcc tgcgattatt gcactcttga ttgaatcaaa gctcccagat
840
attgctgaga acattattta tgttgctgga gatagagtta ttactgatcc tctttgcaag
900
ccattcagca tgggaaggaa tctcctgtgt gtttattcga aaaaacatat gaatgatgac
960
cccgaacttg ctgagatgaa aaagagagca aacacaagaa gcttgaagga gatggctttg
1020
ctattgaggg gtggatcaaa aataatatgg attgctccta gtggtggaag agataggcca
1080
gaccctgtta caaacgaatg gtatccagca ccatttgatg cttccgcgac agacaacatg
1140
aggaggcttg tacaacatgc tggtgtccct ggtcacattt atcctctagc aattttgtgc
1200
catgatatta tgccccctcc cgcccaggtt gagaaaaata tcggggagaa aagagttgta
1260
tcttttcatg gagctggcat atctgtggca cccaaaattg attttcatga ggttgctggt
1320
gctttggagg accctgaggc taagatggta tatacaaagg cactttatga ctctgtaagc
1380
cagcagtaca atgtgctaaa ttctgctata catggcaaac aaggactgaa ggcatcaata
1440
cctagtgttt cattatcaca accatggcag tagcttctct tccaacttta tttttcatat
1500
cttgttgctg tagtcagttt tgcagatgtt tgtttggcag ttacaatcaa atcacaagga
1560
ttacactcac aatctttcca cataccacgc ttgcatgtgg ttagtctatg cagaaagttg
1620
atacaaacaa agtaattctc gaagttacag caaacataac ctgaaggaat ttttttggca
1680
gggttagata attcttttga cacgaatgta cagttgcttt acattgtatt tataccaaat
1740
gttagatcca aatttgttag taatgatagc tttcaagtac tcaattctga ctttttaagg
1800
tcaagtgtta gtagctatcc tagattgctg ctcatcttgc ctttgaagtg gtaatccaat
1860
ttgttgagaa atataataaa tgatgctctg cta
1893
<210> 187
<211> 2016
<212> ДНК
<213> Oryza sativa
<400> 187
gggctggaga tggagatgga gatggagatg gagggtgggt ttggcacaaa tcccgaagcg
60
ctccggcgac cactcccaac ccagtcccca ctagggtaac aacccccttt cggattaggt
120
ttctagaagc ttcttctatg caggcgccgc cgctcgcctc ctcgccgtcg ccggcgtgga
180
ccgccatcct gcccgcgccg gcgaggctct gctgctcccg ccgcggcgcc ctccgcctcg
240
aagccaaggc cgcctggagg ccggcggccc gagggccgcg ggtgccggcc aagggcgccg
300
tcctcgcctc cgaggtggtg ggcccctctc ccctcctcga cgcgcgcaac gagcaagagc
360
tcattttgca tatcagaaag gaagtggaga aagggaagct gcctgcagat gtcgctgcca
420
atctagaaga gctatactac aactacaagg acgcggttat gcagagcagg gatccaaatg
480
cacacgacat cgtgctttca aacatggtgg ccctgttcga ttgtgttctg ctcgatgtag
540
agaatccgtt tacctttccg ccttatcaca aagctgtcag ggaaccattc gactattaca
600
tgtttggtca gaactacatt aggccccttg tagactatag aaattcatat gttggtaata
660
tatccatttt ccaagacatg gaacagaagc tccaacaggg ccataatgtt gttctgatgt
720
ctaaccatca gacagaagca gatccagcaa tcattgcttt gctgcttgaa agaagcaacc
780
catggatcag cgaaaacata gtttatgttg ctggtgatag ggttgttaca gatcctctct
840
gcaagccatt tagtatggga agaaacctca tttgtgtgta ctcaaaaaag catatgaatg
900
attttcctga gctagttgat atgaagagga gggcaaatac tcgtagtctg aaggaaatgg
960
ctttactttt acgtggcggt tcacagataa tttggatagc accaagtggt ggtagagatc
1020
gtccggatcc tttgacagga gaatggcatc cggcaccatt tgatgcatct gcagtggaca
1080
acatgaggag gcttctggag cattctggtg ttcctgggca catatatcca ctctcactgc
1140
tctgctatga ggttatgcct ccaccacaga aggttgagaa agagattggt gagcaaaggg
1200
ttatatcctt ccatggtgta ggcttgtcag taactgaaga gataaagtac agcgacatta
1260
cggttcatac ccaaaatgtc gacgagtgca gagagaaatt ctcagagtca ttgtacaact
1320
cagtcgttga tcagtataat gcgctcaaat ctgctatctt tagaggtcga ggagcagatt
1380
catcggacag tgccatctca ctctcacaac catggcgatg aaactccgct ttctcagttt
1440
tgttctgtct ggatttctca atgaagttac cttcatttct tttcgacaca gcagatgaac
1500
tgctgccgac attgcaattt ttcctggcag aaccttttaa acttcggtat cctaacccat
1560
actaatcatg aaggggaggc tgttactgtc atgcaaatct tgcctagtat gatgatttta
1620
cccagctgaa tcccagccac acatgatgcg ttcgttcatt gtttgcacac aaatattatt
1680
gcgtcatatg agtattcttt gggtcagaac tgcacagcaa cgcggcctgg gcactcaatc
1740
tggcatgttg tctatggggt gcatgcttgt taacagaaga agcccaacat gtgggatttt
1800
gttttttgcg gttaattttt ttcctgtttt ccttttgttc catgtatata tattcgattt
1860
tgatctccag gtttggagat acaatggtca aagtgttatg atagtctctt agtttgttgc
1920
ctcgaagtta tactcgggcg caacatgtct gactgatatt ctgatgatgt tactcgtttc
1980
tgaacttcct gacgccaata tggtgcttgg atgttg
2016
<210> 188
<211> 1888
<212> ДНК
<213> Sorghum bicolor
<400> 188
atgcacgcgc cgccgctggt cgcgttcgca gggggcgcct gccccgccac cgccgcctcg
60
tcctcgccgt cgccctggct ggcctcgccg cgggccgcca tcctcgccgc gccggcgagg
120
ctcctacggt cccgccgcgg ggcacttcgg ctggaagcca aggccgcgtg gagggctgcc
180
ggagggggac ggggcccgag ggtcccggcc aagggcgctg tgctcgcctc ctatatgggc
240
gccgaggagg tggtgggacc ttcgtcgctg ctcgacgagg aagagctcat ttcacatatc
300
agaaaggaac tggataatgg aaaactccct gcagatgttg ccagtaatct ggaggagttg
360
tattataatt acaggaatgc tgttctgcaa aatggagatc ctaatgcata tgagatcatg
420
ctttcaaata tgacggcttt gtttgatcgt gttctactgg atgtacagaa tccatttacc
480
tttccacctt atcacaaagc tgtgcgagaa ccgttcgact attacatgtt tggtcagaac
540
tacattaggc ctctggtaga tttcaggaac tcctatgttg gcaacatttc actttttcat
600
gacatggaag agaagctgca ccagggccac aatgttgttt tgatgtctaa ccatcagaca
660
gaagcagatc cagcaattat ctccttgctt cttgaaaaaa ccaatccatg gattagtgaa
720
aacatagttt atgttgctgg agatagggtt gttatggatc cactttgcaa gccatttagc
780
atgggaagaa atctcatttg cgtgtactcg aaaaagcata tgaatgattt tcctgagcta
840
gttgagatga agaggagatc aaatacccga agtctcaagg aaatggcctt gcttttacgt
900
ggtggctcac agttaatttg gattgcacca agtggtggta gagaccgccc aaatccttca
960
acaggagaat ggtacccggc gccattcgat tcatctgcag tggacaatat gaggaggctt
1020
ctggagcatg ctggcgttcc tgggcacata tatccactat cattgctgtg ctatgaggtt
1080
atgcctccac cacaacaggt tgagaaagag attggtgagc agagggtgat atccttccat
1140
ggagtaggct tgtcagtaac tgaagaaata aaatatgggg atattactgc tcataccaag
1200
aatgctgatg agggaaggga gctattcaca aatactttgt acaactcagt tgttaatcag
1260
tacgatgtgc tcaaatctgc tatctttaga gatcgtggag cagctgtatc aaacaatgtc
1320
atctcattgt cacaaccatg gagatgaatg ttagctttct cagtttgggt ccagatttat
1380
tactgaagtt accttttcag aagagcaggt gaactgccat tgtgcaattt cactggagaa
1440
actcttgaac tttaatcttt ttgataccac tcgactttat cagtcatggt ggagcctgtc
1500
attgtcatgc agatccttgc taagaagtct gtggacaact gttggttggt caagggtgac
1560
tggtgattct gcacataggg atcctcgtaa ctgttgcatg cggtcgtccg caaattactg
1620
gttgctcagc aacgtgctgg ttgggcactg aggaatccgt caggttgcat cctttttgcc
1680
ttgacgtcaa tttgtgtagt tgaaggttga agtgataaat tgttttatct tgtcttgtca
1740
tcatgtatat aggctcgagt cttttttggc tccacatttt tttggagata taaaagcagc
1800
aggagttatg acatgccctc agtcggccct ccttgttgaa accctttgga tgtaacctgt
1860
ctatttctta tatatactca ctgaaagt
1888
<210> 189
<211> 2046
<212> ДНК
<213> Zea mays
<400> 189
gaggattcat ctcgtgtcga cgacgccttc gccctctccg agtctccgtc cgtcttccgc
60
gtcctccgca gctggactcg tgcgctattc cccaggacgc tactcccact agggttttcg
120
gattaggttt ctagaacctt ccaccgccgc ctctccatgc acgcgccgcc gctggtcacg
180
ttcgcagggg gcgcctgccc caccaccgcc tccgcttcgc cgtcgccctg gttggcctcg
240
ccgcgggacg ccatctttgc cgcgccggcg aggcccctac ggtcccgccg cgggacactc
300
cggctggaag ctaaggccgc gtggagggct gccggagggg gacggggccc gcgggtcccg
360
gccaagggcg ctgtgctcgc ctcctatatg ggcgccgagg aggtggtggg accatcgtcg
420
ctgcttgacg aggaagagtt catttctcac atcagaaagg aactggataa tggaaaactt
480
cctgcagatg ttgccagtaa cctggaggag ctgtattata attacaggaa tgcggttctg
540
caaaatggag atccaaatgc atatgaggtc atgctttcaa atatgatgac cttgtttgat
600
cgtgttctac tggatgtaca gaatccattt aactttccac cttatcataa agctttgcga
660
gaaccgttcg actattacat gtttggtcag aactacatta ggcctctggt agatttcagg
720
aactcctatg ttggcaacat ttcccttttc catgatatcg aagagaatct ccaccagggc
780
cacaatgttg ttttgatgtc taaccatcag tcagaagcag atccagcaat tattgccttg
840
cttcttgaaa aaaccaatcc ttggattagt gaaaacatag tttatgttgc tggcgatagg
900
gttgttaccg atccgctttg caagccattt agcatgggaa gaaatctcat ttgcgtgtac
960
tcgaaaaagc atatgaatga tttccctgag ctaattgaga tgaagaggag atcaaatact
1020
cgaagtctca aggaaatggc attgctttta cgtggtggtt cacagttaat ttggattgca
1080
ccgagtggtg gtagagaccg cccaaatccc tcatcaggag aatggtaccc ggcaccattc
1140
gattcatctg cagtggacaa tatgaggagg cttctggagc atgctggtgt tcctgggcac
1200
atatatccac tatcattgct gtgctatgag gttatgcctc caccacaaca ggttgagaag
1260
gagattggtg agcagagggt gatatccttc catggagcag gcttgtcagt aactgaagaa
1320
ataaactatg gagacattac tgctcatacc aagaatgctg atgagggaag ggagctattc
1380
acaaatacct tgtacaactc agttgttaac cagtacaatg tgctcaaatc tgctatcttt
1440
agagatcgtg gagcagctgt atcaaacaat gtcatctcac tgtcacaacc atggcgatga
1500
atgtccagtt tcgttactga agttaccttt tcaaaagagc aggtgaacta tcattgtgca
1560
attttgctgg gagaaactct tgaacttaaa tctttttgat atcactagac ttcatcaatc
1620
atggtggagc ctgttattgt catgcagatg ctcgctaaga agtctgcaac tgttggttgg
1680
tcaagggtga ttggcgattt tgcacatacg gatccgcagt tactgctgcc tcaggttgct
1740
cagcaatgtg ctgctggttg ggcaccgagg aatccatcag gttgcatcct ttttgcccgg
1800
acgtcgattt gtgcagttga agtggtaaac gtttttttat cttgtttcgt catcatgtat
1860
atacgtaggc ttgagtcatt gttggctcca cattttttgg agatataaat gcggtaggag
1920
ttatgagatg tccacagtcg gccctccctt gttgaattcc tttggatgta tcctctctct
1980
ttcgtacatg cactcactga aagtcaatgc aaatatctcg tgtttctagt aaaaaaaaaa
2040
aaaaaa
2046
<210> 190
<211> 1994
<212> ДНК
<213> Hordeum vulgare
<400> 190
gagaaaccaa gaagcgagaa tcggcaccgt tcccgtcgcc gcctcgtcgc ctccgtcgcc
60
cgtcttcctt ttccgccgat tcgtgcccac caccaccact ctccttccca ctggctaggg
120
ttttcggttt ctagaacctc acgcccgccc gctccatgca agccccgccg ctcgccgcgc
180
tcgccggagg cgcctgggcc tctcaccgac ctgccatact agcggcgccg gcgggcctcc
240
gccgtcccag gcgctgcgcc ctccggctgc ccgcgtggag ggcggccgga ggcggccggg
300
ccccgcggct accggtcaag ggcgccgtgc tcgcctccga cacgggggcg gacgaggagg
360
tcgcggggcc atcgcccctg ctcgacgtgc gcagcgagca agagttcgtt ctacgcgtca
420
ggaaggaagt ggagagaggg aagttgcgtc cagatgttgc tgacaacttt gaaaacctgt
480
actgcaatta caagaatgcg gtgctacaaa atggggatcc aaatgcatat cagatcatgc
540
tttccaacat gatggattta tttgaccgcg ttctgctaga tgcagagaat ccatttacgt
600
ttcagcctta tcacaaggcc atcagagaac cgtttgacta ttacactttc ggtcagaact
660
acattaggcc actggtagat tttaggaact cttatgtcgg taacatttct gtattcagtg
720
atatggagaa gcagctccgg cagggtcata atgttgttct gatgtctaat catcagacag
780
aagcggatcc agcagttatt gccttgtcac ttgaaagaag caatccgtgg attagcgaga
840
acatagttta tgttgctggg gatagggttc ttacagatcc tctttgcaag ccatttagca
900
tgggaagaaa cctcctttgt gtgtactcaa aaaagcatat gaatgatttt cctgagctaa
960
ttgagatgaa gaggagggca aacactcgaa gtctcaagga aatggctttg cttttacgtg
1020
ggggttcaca tataatttgg atagctccga gtggcggtag agaccgtcct gaccccttga
1080
ctggagaatg gcacccggcg ccatttgatg catctgcagt ggataatatg aggaggcttc
1140
tggagcattc tggcgttcct gggcacatat atccattatc attgctatgc tatgagatta
1200
tgcctccacc acaacagatt gagaaagaga ttggtgagca aagggtgata tccttccatg
1260
gtgtaggctt gtcagtagct gaagaaataa agtatgggga tgttactgct caatctcaga
1320
atgctgatga ggcaaggggg aacttctcag aggctctgta cagttcagtt gttgatcaat
1380
ataatgtcct caagtctgct atctttagag accgtggagc agtttcgtcg aaccctgcca
1440
tctcactctc gcaaccatgg cggtgaaact aagctttctc aggcctggat ttctcatttc
1500
ttttcgacag agcagatgaa ctgctatagt gcaacgttgt ggttttttgc tgggatggcc
1560
ttaaactttg atgtcgtcac agttaggatg aggccctgca gatcctgtaa gttgttgaag
1620
tcgcgggaag gaaaaaccgt gtgatatgct gctacaccgt gttcatgtag tgacaggaag
1680
tctgcggctg ttgtcaggtc taaatcctaa atagcacggc ggaacccagc agcagatgat
1740
gcatgtgttc atcttttgtg aacagctact gctgtatcag atggctatca tctgggccag
1800
attggtccag caaatacaga ttggcccctg gatcctggca gtcgtctgga tcaaaatgct
1860
gatatttctt tttgtgctcg tcttattttt ttgattagtt ttgtgtacat attaattctt
1920
ttgctccaaa atttggagac acatgacatg atatatacag agcagagccc aatatgtgtc
1980
gccttcctgt taac
1994
<210> 191
<211> 1936
<212> ДНК
<213> Physcomitrella patens
<400> 191
ggacgagcgg agtggagagc tatggcggca gcagctggtt ccgctggcgt ggtatgttgg
60
tctagggcag agaagcagca tgccccggtc agggggggtg gaactagtgt taccagtagt
120
accagtggca gcggccatgc gtcgttgaaa gggagcttcg atcggctcca aggtaaccgc
180
cttctgccgc aagccttgac tatgccgtcg ctgtttcggg cgaaacgcaa tggcagaagg
240
acgccgggga atgccgtgac caatttcggg aaatctgaat tccatcgtga aattagtggg
300
agtacgcggg cgaccacgca ggtggctgaa gccaccacag ctggtcttag ggagaccatt
360
gaggaccgcg ctattatcga cggtcattct cacagttttg aaggaattca atcggaagaa
420
gagttgatgc aggtaattga aaaggaggtg gaatccggtc ggctgccgaa gcgtgctggc
480
gcgggaatgg tagagttgta tcgcaattat cgagatgctg tagtgagcag tggcgtagaa
540
aatgcgatgg atattgttgt gaaagtcatg tcaactgtgt tggaccggat tcttctgcag
600
ttcgaggagc cattcacatt tggatcgcac cacaagagaa tggtggagcc gtatgattac
660
tacacatttg gtcagaacta tgtgcgtcct ctcctagatt tcaggaactc ttaccttggg
720
aacttaaaga tctttgacca gatagagaag aacctgaaag aggggcacaa cgtcattttt
780
ctatccaatc accagactga ggcagatcct gctgttatgg cgctgttgct tgagcactct
840
cacccctatt tggcagagaa cttgacctat gtggctggag acagggttgt gctggatcca
900
ttctgcaaac cttttagtat gggcaggaat ctcttgtgcg tgtattcaaa aaagcacatt
960
cacgatgtac cggaccttgc tgaaatgaaa atcaaagcta atgcgaagac tttgagacag
1020
atgacgatcc tgctgaggca gggaggtcaa ttattatggg tagcacccag tggtggacgc
1080
gatcgccctg atcctgagac caacgaatgg gttcctgcac attttgactc gtctgctgtg
1140
gagaatatga agcgactatc tgacattgtc cgagtacctg ctcatttaca tgccctatca
1200
ttactatgtt ttgagattat gccacctcct gtccaggtac aaaaggagct aggagagcga
1260
agagcagtag gatttagcgg agttggtcta gccgtttccg agcaactaga ttatgattcc
1320
attgcgaagt tagtcgacga ttccaaaaat gcgaaggatg ccttttcgga tgcggcatgg
1380
agcgaagtca atgatatgta taacgtgtta aaagaagcaa tttatggtga ccaaggttgt
1440
gctgttagca cagattcctt gagactggaa cagccctggt ttgatggaag caggcgaact
1500
gattgaaaat aggtcatttg aagttttatg taaaagtatg aagcatcctt attgcttttt
1560
acgctgtcta agctccaagg atgtaagaat tcagcagcgt gtataatggc tacattgtca
1620
tgtgatattc tttctgattc gtgcgacacg atggccatgc ctgctcaatc cttgtcacca
1680
ggcgtctcag taggaaacgg tggtactgat tgctgtctgt ccgacttgat ttagtagctc
1740
ggattctgcg tactggataa cttggtctgg taatagggac cgatcctatc ggtgaggagt
1800
ttgtgatata gatcaatact gcactttgtt acaatcggaa tagatgcatt cattattcat
1860
ccaagccaac acatcctgag ttggagcata agttgaagca ctcctcaact tcattgaaag
1920
gagatttctc actacg
1936
<210> 192
<211> 2260
<212> ДНК
<213> Chlamydomonas reinhardtii
<400> 192
gaagaatgct gcacgcgact cagcagcgcg cggtcgctgg ccgtcgcccg ttctcgggtg
60
cgcgcgcgtc gaaccgcgtt gttgctcacg cggctgcgac cgtcgccacc agtctgccga
120
ccgttgacgt ccagttccac cagcctaagc tggcgggcgt gaccaacgag cagcagttca
180
aggcggtaat caaggggctg gtcgctcagg gcaagttccc tccgcagctg gagcccgctt
240
gggattactt ctatgacaac tacaagaagg ctgtcaccag cagtggcgtc gctggggccg
300
atgagaagct tgtcacccag gtgcaagcca gcattctgga caatgtcctg aaccaggcgg
360
tgaaccccta caccttcccc tctttccaca cccgcctaat tgagccctac aactactatg
420
acttcggtca gcgctacgtc gcgaccctca tcgacttcca gaactccgtg ctgggtttcc
480
gcgagcgttt cgaccgcgtt caggagctgc tggaccagaa gcacaacgtt gttatcctcg
540
cgaaccacca gacggaggcc gaccccggtg tgtttgccca tatgctggcg aagacgcacc
600
ctaagctggc gacggatgtg atctacgtcg ctggcgaccg cgttgtcacc gatccgatgt
660
gcaagccctt ctccatgggc cgcaacctct tctgcgtgca ctccaagaag cacatggacg
720
acgctccgga gctgaaggcc gcaaagatgg agaccaaccg caagacgctg gtcgccatgc
780
aacgcaagct gaacgagggc ggcacgctca tgtggatcgc ccccagcggc ggccgcgacc
840
gccccaacgc caacgacgag tgggtgcccg ataactttga tcccgccgcc gtggagctga
900
tgcgcaacct ggtgcagcgc gccaagcagc cgggccacct gatgcccatg tccatgttca
960
gctaccccat gatgccgccg cccaagaccg tggacaagtc cattggcgag cgccgcctca
1020
cggccttcac gggcgtgggc atctccctgt gcgaggagct ggacgtggcg gccatcatcg
1080
cggccagcgg ctcggaggag aaggagcaga aggctctggc caaggccgcg cacgacgcgg
1140
tgaaggagtc gtacgcggtg ctgtccaagg ccatccagga tcccgccttc cgcgccaccc
1200
gcaaggagtt cacacagccc tggatggcgt aaggaggcgg gagcagcagc ggcagtggcg
1260
gcagcgacag cagtggcgga tcttgggggc acgaggcagc agctagcaca cgcggggccg
1320
gtggcggcag cggacgggag aggtgctggt gcggatgctg gtgccaggag cagtgcgtct
1380
atgcctggcg gcggcgccga ccggtgctga agctgttgtc ggcagcagca gcgggaactt
1440
gcgctggcga tggctgaagg tgatgtggct ggccgtacag caaatgctgc tgtcacgcat
1500
tgtcagcggc ggccgctggg tccgctggga tgtgaggagt gcgagatcag cataggccag
1560
gcgggtgggt tgcggtgccg ctgccgacac gtcgcacagg aaggagcagg aagggtgcgg
1620
ctgagcacca gggtcacttg gggcctgtgt tagcgtgacc gggcctgaaa ggggtatttg
1680
tctggggagc agctcgacgc acattcgtgg gattgcttag gaaggagcgt tgggatggct
1740
gtgccgcgcg gtgtgcccag cacgttgact ggctctgacc cgggtcaacc aatgcgttgc
1800
ggccgttgca acgatgcttc taatcagcgt gagatggagt gtacgaatgc aggtatgacg
1860
atgaccatag acgagtgtga gcgtgtgtat ctgtggattg gacacggttc attcaatcca
1920
ttcgtaccgg acatatgact atacaagacc gtgtggagtg tgtttgtgcg ctcaagactg
1980
gacgaagtgg gcgctcccga tgtggagcgc ggtgctcgcg ggttgtgtgt cttgccgcaa
2040
cgcaagcagc gtggcgtggt gtggatgatt cttgcattat gactggggtt ttaccggcgg
2100
cccagagcgt gtgtggagca aaagcaaaaa caggaacaag gagccgtgtg cacaattcgc
2160
gcggatggtg gtggctgggg atctgacagg agagtgccaa cggcggccgg gtgctagtgc
2220
ttgaacatca tgtgatattg tgattgtaca aatggactgc
2260
<210> 193
<211> 362
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 193
Met Leu Lys Leu Ser Cys Asn Val Thr Asp Ser Lys Leu Gln Arg Ser
1 5 10 15
Leu Leu Phe Phe Ser His Ser Tyr Arg Ser Asp Pro Val Asn Phe Ile
20 25 30
Arg Arg Arg Ile Val Ser Cys Ser Gln Thr Lys Lys Thr Gly Leu Val
35 40 45
Pro Leu Arg Ala Val Val Ser Ala Asp Gln Gly Ser Val Val Gln Gly
50 55 60
Leu Ala Thr Leu Ala Asp Gln Leu Arg Leu Gly Ser Leu Thr Glu Asp
65 70 75 80
Gly Leu Ser Tyr Lys Glu Lys Phe Val Val Arg Ser Tyr Glu Val Gly
85 90 95
Ser Asn Lys Thr Ala Thr Val Glu Thr Ile Ala Asn Leu Leu Gln Glu
100 105 110
Val Gly Cys Asn His Ala Gln Ser Val Gly Phe Ser Thr Asp Gly Phe
115 120 125
Ala Thr Thr Thr Thr Met Arg Lys Leu His Leu Ile Trp Val Thr Ala
130 135 140
Arg Met His Ile Glu Ile Tyr Lys Tyr Pro Ala Trp Gly Asp Val Val
145 150 155 160
Glu Ile Glu Thr Trp Cys Gln Ser Glu Gly Arg Ile Gly Thr Arg Arg
165 170 175
Asp Trp Ile Leu Lys Asp Ser Val Thr Gly Glu Val Thr Gly Arg Ala
180 185 190
Thr Ser Lys Trp Val Met Met Asn Gln Asp Thr Arg Arg Leu Gln Lys
195 200 205
Val Ser Asp Asp Val Arg Asp Glu Tyr Leu Val Phe Cys Pro Gln Glu
210 215 220
Pro Arg Leu Ala Phe Pro Glu Glu Asn Asn Arg Ser Leu Lys Lys Ile
225 230 235 240
Pro Lys Leu Glu Asp Pro Ala Gln Tyr Ser Met Ile Gly Leu Lys Pro
245 250 255
Arg Arg Ala Asp Leu Asp Met Asn Gln His Val Asn Asn Val Thr Tyr
260 265 270
Ile Gly Trp Val Leu Glu Ser Ile Pro Gln Glu Ile Val Asp Thr His
275 280 285
Glu Leu Gln Val Ile Thr Leu Asp Tyr Arg Arg Glu Cys Gln Gln Asp
290 295 300
Asp Val Val Asp Ser Leu Thr Thr Thr Thr Ser Glu Ile Gly Gly Thr
305 310 315 320
Asn Gly Ser Ala Thr Ser Gly Thr Gln Gly His Asn Asp Ser Gln Phe
325 330 335
Leu His Leu Leu Arg Leu Ser Gly Asp Gly Gln Glu Ile Asn Arg Gly
340 345 350
Thr Thr Leu Trp Arg Lys Lys Pro Ser Ser
355 360
<210> 194
<211> 367
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 194
Met Leu Lys Leu Ser Cys Asn Val Thr Asp His Ile His Asn Leu Phe
1 5 10 15
Ser Asn Ser Arg Arg Ile Phe Val Pro Val His Arg Gln Thr Arg Pro
20 25 30
Ile Ser Cys Phe Gln Leu Lys Lys Glu Pro Leu Arg Ala Ile Leu Ser
35 40 45
Ala Asp His Gly Asn Ser Ser Val Arg Val Ala Asp Thr Val Ser Gly
50 55 60
Thr Ser Pro Ala Asp Arg Leu Arg Phe Gly Arg Leu Met Glu Asp Gly
65 70 75 80
Phe Ser Tyr Lys Glu Lys Phe Ile Val Arg Ser Tyr Glu Val Gly Ile
85 90 95
Asn Lys Thr Ala Thr Ile Glu Thr Ile Ala Asn Leu Leu Gln Glu Val
100 105 110
Ala Cys Asn His Val Gln Asn Val Gly Phe Ser Thr Asp Gly Phe Ala
115 120 125
Thr Thr Leu Thr Met Arg Lys Leu His Leu Ile Trp Val Thr Ala Arg
130 135 140
Met His Ile Glu Ile Tyr Lys Tyr Pro Ala Trp Ser Asp Val Val Glu
145 150 155 160
Ile Glu Thr Trp Cys Gln Ser Glu Gly Arg Ile Gly Thr Arg Arg Asp
165 170 175
Trp Ile Leu Lys Asp Cys Ala Thr Gly Glu Val Ile Gly Arg Ala Thr
180 185 190
Ser Lys Trp Val Met Met Asn Gln Asp Thr Arg Arg Leu Gln Arg Val
195 200 205
Thr Asp Glu Val Arg Asp Glu Tyr Leu Val Phe Cys Pro Pro Glu Pro
210 215 220
Arg Leu Ala Phe Pro Glu Glu Asn Asn Ser Ser Leu Lys Lys Ile Pro
225 230 235 240
Lys Leu Glu Asp Pro Ala Gln Tyr Ser Met Leu Gly Leu Lys Pro Arg
245 250 255
Arg Ala Asp Leu Asp Met Asn Gln His Val Asn Asn Val Thr Tyr Ile
260 265 270
Gly Trp Val Leu Glu Ser Ile Pro Gln Glu Ile Ile Asp Thr His Glu
275 280 285
Leu Lys Val Ile Thr Leu Asp Tyr Arg Arg Glu Cys Gln Gln Asp Asp
290 295 300
Ile Val Asp Ser Leu Thr Thr Ser Glu Thr Pro Asn Glu Val Val Ser
305 310 315 320
Lys Leu Thr Gly Thr Asn Gly Ser Thr Thr Ser Ser Lys Arg Glu His
325 330 335
Asn Glu Ser His Phe Leu His Ile Leu Arg Leu Ser Glu Asn Gly Gln
340 345 350
Glu Ile Asn Arg Gly Arg Thr Gln Trp Arg Lys Lys Ser Ser Arg
355 360 365
<210> 195
<211> 412
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 195
Met Val Ala Thr Ser Ala Thr Ser Ser Phe Phe Pro Val Pro Ser Ser
1 5 10 15
Ser Leu Asp Pro Asn Gly Lys Gly Asn Lys Ile Gly Ser Thr Asn Leu
20 25 30
Ala Gly Leu Asn Ser Ala Pro Asn Ser Gly Arg Met Lys Val Lys Pro
35 40 45
Asn Ala Gln Ala Pro Pro Lys Ile Asn Gly Lys Lys Val Gly Leu Pro
50 55 60
Gly Ser Val Asp Ile Val Arg Thr Asp Thr Glu Thr Ser Ser His Pro
65 70 75 80
Ala Pro Arg Thr Phe Ile Asn Gln Leu Pro Asp Trp Ser Met Leu Leu
85 90 95
Ala Ala Ile Thr Thr Ile Phe Leu Ala Ala Glu Lys Gln Trp Met Met
100 105 110
Leu Asp Trp Lys Pro Arg Arg Ser Asp Met Leu Val Asp Pro Phe Gly
115 120 125
Ile Gly Arg Ile Val Gln Asp Gly Leu Val Phe Arg Gln Asn Phe Ser
130 135 140
Ile Arg Ser Tyr Glu Ile Gly Ala Asp Arg Ser Ala Ser Ile Glu Thr
145 150 155 160
Val Met Asn His Leu Gln Glu Thr Ala Leu Asn His Val Lys Thr Ala
165 170 175
Gly Leu Leu Gly Asp Gly Phe Gly Ser Thr Pro Glu Met Phe Lys Lys
180 185 190
Asn Leu Ile Trp Val Val Thr Arg Met Gln Val Val Val Asp Lys Tyr
195 200 205
Pro Thr Trp Gly Asp Val Val Glu Val Asp Thr Trp Val Ser Gln Ser
210 215 220
Gly Lys Asn Gly Met Arg Arg Asp Trp Leu Val Arg Asp Cys Asn Thr
225 230 235 240
Gly Glu Thr Leu Thr Arg Ala Ser Ser Val Trp Val Met Met Asn Lys
245 250 255
Leu Thr Arg Arg Leu Ser Lys Ile Pro Glu Glu Val Arg Gly Glu Ile
260 265 270
Glu Pro Tyr Phe Val Asn Ser Asp Pro Val Leu Ala Glu Asp Ser Arg
275 280 285
Lys Leu Thr Lys Ile Asp Asp Lys Thr Ala Asp Tyr Val Arg Ser Gly
290 295 300
Leu Thr Pro Arg Trp Ser Asp Leu Asp Val Asn Gln His Val Asn Asn
305 310 315 320
Val Lys Tyr Ile Gly Trp Ile Leu Glu Ser Ala Pro Val Gly Ile Met
325 330 335
Glu Arg Gln Lys Leu Lys Ser Met Thr Leu Glu Tyr Arg Arg Glu Cys
340 345 350
Gly Arg Asp Ser Val Leu Gln Ser Leu Thr Ala Val Thr Gly Cys Asp
355 360 365
Ile Gly Asn Leu Ala Thr Ala Gly Asp Val Glu Cys Gln His Leu Leu
370 375 380
Arg Leu Gln Asp Gly Ala Glu Val Val Arg Gly Arg Thr Glu Trp Ser
385 390 395 400
Ser Lys Thr Pro Thr Thr Thr Trp Gly Thr Ala Pro
405 410
<210> 196
<211> 345
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 196
Met Phe Ile Ala Val Glu Val Ser Pro Val Met Glu Asp Ile Thr Arg
1 5 10 15
Gln Ser Lys Lys Thr Ser Val Glu Asn Glu Thr Gly Asp Asp Gln Ser
20 25 30
Ala Thr Ser Val Val Leu Lys Ala Lys Arg Lys Arg Arg Ser Gln Pro
35 40 45
Arg Asp Ala Pro Pro Gln Arg Ser Ser Val His Arg Gly Val Thr Arg
50 55 60
His Arg Trp Thr Gly Arg Tyr Glu Ala His Leu Trp Asp Lys Asn Ser
65 70 75 80
Trp Asn Glu Thr Gln Thr Lys Lys Gly Arg Gln Val Tyr Leu Gly Ala
85 90 95
Tyr Asp Glu Glu Asp Ala Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu
100 105 110
Lys Tyr Trp Gly Arg Asp Thr Ile Leu Asn Phe Pro Leu Cys Asn Tyr
115 120 125
Glu Glu Asp Ile Lys Glu Met Glu Ser Gln Ser Lys Glu Glu Tyr Ile
130 135 140
Gly Ser Leu Arg Arg Lys Ser Ser Gly Phe Ser Arg Gly Val Ser Lys
145 150 155 160
Tyr Arg Gly Val Ala Lys His His His Asn Gly Arg Trp Glu Ala Arg
165 170 175
Ile Gly Arg Val Phe Gly Asn Lys Tyr Leu Tyr Leu Gly Thr Tyr Ala
180 185 190
Thr Gln Glu Glu Ala Ala Ile Ala Tyr Asp Ile Ala Ala Ile Glu Tyr
195 200 205
Arg Gly Leu Asn Ala Val Thr Asn Phe Asp Ile Ser Arg Tyr Leu Lys
210 215 220
Leu Pro Val Pro Glu Asn Pro Ile Asp Thr Ala Asn Asn Leu Leu Glu
225 230 235 240
Ser Pro His Ser Asp Leu Ser Pro Phe Ile Lys Pro Asn His Glu Ser
245 250 255
Asp Leu Ser Gln Ser Gln Ser Ser Ser Glu Asp Asn Asp Asp Arg Lys
260 265 270
Thr Lys Leu Leu Lys Ser Ser Pro Leu Val Ala Glu Glu Val Ile Gly
275 280 285
Pro Ser Thr Pro Pro Glu Ile Ala Pro Pro Arg Arg Ser Phe Pro Glu
290 295 300
Asp Ile Gln Thr Tyr Phe Gly Cys Gln Asn Ser Gly Lys Leu Thr Ala
305 310 315 320
Glu Glu Asp Asp Val Ile Phe Gly Asp Leu Asp Ser Phe Leu Thr Pro
325 330 335
Asp Phe Tyr Ser Glu Leu Asn Asp Cys
340 345
<210> 197
<211> 303
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 197
Met Ala Lys Val Ser Gly Arg Ser Lys Lys Thr Ile Val Asp Asp Glu
1 5 10 15
Ile Ser Asp Lys Thr Ala Ser Ala Ser Glu Ser Ala Ser Ile Ala Leu
20 25 30
Thr Ser Lys Arg Lys Arg Lys Ser Pro Pro Arg Asn Ala Pro Leu Gln
35 40 45
Arg Ser Ser Pro Tyr Arg Gly Val Thr Arg His Arg Trp Thr Gly Arg
50 55 60
Tyr Glu Ala His Leu Trp Asp Lys Asn Ser Trp Asn Asp Thr Gln Thr
65 70 75 80
Lys Lys Gly Arg Gln Val Tyr Leu Gly Ala Tyr Asp Glu Glu Glu Ala
85 90 95
Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Arg Asp
100 105 110
Thr Leu Leu Asn Phe Pro Leu Pro Ser Tyr Asp Glu Asp Val Lys Glu
115 120 125
Met Glu Gly Gln Ser Lys Glu Glu Tyr Ile Gly Ser Leu Arg Arg Lys
130 135 140
Ser Ser Gly Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly Val Ala Arg
145 150 155 160
His His His Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg Val Phe Ala
165 170 175
Thr Gln Glu Glu Ala Ala Ile Ala Tyr Asp Ile Ala Ala Ile Glu Tyr
180 185 190
Arg Gly Leu Asn Ala Val Thr Asn Phe Asp Val Ser Arg Tyr Leu Asn
195 200 205
Pro Asn Ala Ala Ala Asp Lys Ala Asp Ser Asp Ser Lys Pro Ile Arg
210 215 220
Ser Pro Ser Arg Glu Pro Glu Ser Ser Asp Asp Asn Lys Ser Pro Lys
225 230 235 240
Ser Glu Glu Val Ile Glu Pro Ser Thr Ser Pro Glu Val Ile Pro Thr
245 250 255
Arg Arg Ser Phe Pro Asp Asp Ile Gln Thr Tyr Phe Gly Cys Gln Asp
260 265 270
Ser Gly Lys Leu Ala Thr Glu Glu Asp Val Ile Phe Asp Cys Phe Asn
275 280 285
Ser Tyr Ile Asn Pro Gly Phe Tyr Asn Glu Phe Asp Tyr Gly Pro
290 295 300
<210> 198
<211> 445
<212> БЕЛОК
<213> Avena sativa
<400> 198
Met Lys Arg Ser Pro Pro Pro Ala Pro Pro Ala Ala Pro Pro Pro Pro
1 5 10 15
Gln Pro Ser Pro Ser Ser Ser Ser Pro Ala Cys Ser Pro Ser Pro Ser
20 25 30
Ser Ser Ser Cys Pro Ser Ser Ser Asp Ser Ser Ser Ile Val Ile Pro
35 40 45
Arg Lys Arg Ala Arg Thr Gln Lys Ala Ala Ser Gly Lys Pro Lys Ala
50 55 60
Lys Ala Ser Ala Lys Arg Pro Lys Lys Asp Ala Ser Arg Ser Ser Lys
65 70 75 80
Glu Thr Asp Ala Asn Gly Ala Ala Ala Ala Ala Gly Lys Arg Ser Ser
85 90 95
Ile Tyr Arg Gly Val Thr Arg His Arg Trp Thr Gly Arg Phe Glu Ala
100 105 110
His Leu Trp Asp Lys Asn Cys Phe Thr Ser Val Gln Asn Lys Lys Lys
115 120 125
Gly Arg Gln Val Tyr Leu Gly Ala Tyr Asp Thr Glu Asp Ala Ala Ala
130 135 140
Arg Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Ser Glu Thr Ile
145 150 155 160
Leu Asn Phe Ser Val Glu Asp Tyr Ala Lys Glu Met Pro Glu Met Glu
165 170 175
Ala Val Ser Arg Glu Glu Tyr Leu Ala Ala Leu Arg Arg Arg Ser Ser
180 185 190
Gly Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly Val Ala Arg His His
195 200 205
His Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg Val Leu Gly Asn Lys
210 215 220
Tyr Leu Tyr Leu Gly Thr Phe Asp Thr Gln Glu Glu Ala Ala Lys Ala
225 230 235 240
Tyr Asp Leu Ala Ala Ile Glu Tyr Arg Gly Ala Asn Ala Val Thr Asn
245 250 255
Phe Asp Ile Ser Cys Tyr Leu Asp Gln Pro Gln Leu Leu Ala Gln Leu
260 265 270
Gln Gln Gly Pro Gln Val Val Pro Ala Leu Gln Glu Glu Leu Gln His
275 280 285
Asp Val Gln His Asp Leu Gln Asn Asp Asn Ala Val Gln Glu Leu Asn
290 295 300
Ser Gly Glu Val Gln Met Pro Gly Ala Met Asp Glu Pro Ile Ala Leu
305 310 315 320
Asp Asp Ser Thr Glu Cys Ile Asn Thr Pro Phe Glu Phe Asp Phe Ser
325 330 335
Val Glu Glu Asn Leu Trp Ser Pro Cys Met Asp Tyr Glu Leu Asp Ala
340 345 350
Ile Leu Gly Asn Asn Thr Ser Asn Ser Ala Asn Met Asn Glu Trp Phe
355 360 365
Asn Asp Ser Thr Phe Glu Ser Asn Ile Gly Cys Leu Phe Glu Gly Cys
370 375 380
Ser Asn Ile Asp Asp Cys Ser Ser Ser Lys His Cys Ala Asp Leu Ala
385 390 395 400
Ala Phe Asp Phe Phe Lys Glu Gly Asp Asp Asn Asp Phe Ser Asn Met
405 410 415
Glu Met Glu Ile Thr Pro Gln Ala Asn Asp Val Ser Cys Pro Pro Asn
420 425 430
Asp Val Ser Cys Pro Pro Lys Met Ile Thr Val Cys Asn
435 440 445
<210> 199
<211> 420
<212> БЕЛОК
<213> Sorghum bicolor
<400> 199
Met Asp Met Glu Arg Ser Gln Gln Gln Lys Ser Pro Thr Glu Ser Pro
1 5 10 15
Pro Pro Pro Ser Pro Ser Ser Ser Ser Ser Ser Val Ser Ala Asp Thr
20 25 30
Val Leu Pro Pro Pro Gly Lys Arg Arg Arg Ala Ala Thr Thr Ala Lys
35 40 45
Ala Lys Ala Gly Ala Lys Pro Lys Arg Ala Arg Lys Asp Ala Ala Ala
50 55 60
Ala Ala Asp Pro Pro Pro Pro Pro Ala Ala Ala Ala Ala Gly Lys Arg
65 70 75 80
Ser Ser Val Tyr Arg Gly Val Thr Arg His Arg Trp Thr Gly Arg Phe
85 90 95
Glu Ala His Leu Trp Asp Lys His Cys Leu Ala Ala Leu His Asn Lys
100 105 110
Lys Lys Gly Arg Gln Val Tyr Leu Gly Ala Tyr Asp Ser Glu Glu Ala
115 120 125
Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Pro Glu
130 135 140
Thr Leu Leu Asn Phe Pro Val Glu Asp Tyr Ser Ser Glu Met Pro Glu
145 150 155 160
Met Glu Gly Val Ser Arg Glu Glu Tyr Leu Ala Ser Leu Arg Arg Arg
165 170 175
Ser Ser Gly Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly Val Ala Arg
180 185 190
His His His Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg Val Phe Gly
195 200 205
Asn Lys Tyr Leu Tyr Leu Gly Thr Phe Asp Thr Gln Glu Glu Ala Ala
210 215 220
Lys Ala Tyr Asp Leu Ala Ala Ile Glu Tyr Arg Gly Val Asn Ala Val
225 230 235 240
Thr Asn Phe Asp Ile Ser Cys Tyr Leu Asp His Pro Leu Phe Leu Ala
245 250 255
Gln Leu Gln Gln Glu Pro Gln Val Val Pro Ala Leu Asn Gln Glu Ala
260 265 270
Gln Pro Asp Gln Ser Glu Thr Glu Thr Ile Ala Gln Glu Ser Val Ser
275 280 285
Ser Glu Ala Lys Thr Pro Asp Asp Asn Ala Glu Pro Asp Asp Asn Ala
290 295 300
Glu Pro Asp Asp Ile Ala Glu Pro Leu Ile Thr Val Asp Asp Ser Ile
305 310 315 320
Glu Glu Ser Leu Trp Ser Pro Cys Met Asp Tyr Glu Leu Asp Thr Met
325 330 335
Ser Arg Ser Asn Phe Gly Ser Ser Ile Asn Leu Ser Glu Trp Phe Asn
340 345 350
Asp Ala Asp Phe Asp Ser Asn Ile Gly Cys Leu Phe Asp Gly Cys Ser
355 360 365
Ala Val Asp Glu Gly Gly Lys Asp Gly Val Gly Leu Ala Asp Phe Ser
370 375 380
Leu Leu Glu Asp Phe Ser Leu Phe Glu Ala Gly Asp Gly Gln Leu Lys
385 390 395 400
Asp Val Leu Ser Asp Met Glu Glu Gly Ile Gln Pro Pro Thr Met Ile
405 410 415
Ser Val Cys Asn
420
<210> 200
<211> 395
<212> БЕЛОК
<213> Zea mays
<400> 200
Met Glu Arg Ser Gln Arg Gln Ser Pro Pro Pro Pro Ser Pro Ser Ser
1 5 10 15
Ser Ser Ser Ser Val Ser Ala Asp Thr Val Leu Val Pro Pro Gly Lys
20 25 30
Arg Arg Arg Ala Ala Thr Ala Lys Ala Gly Ala Glu Pro Asn Lys Arg
35 40 45
Ile Arg Lys Asp Pro Ala Ala Ala Ala Ala Gly Lys Arg Ser Ser Val
50 55 60
Tyr Arg Gly Val Thr Arg His Arg Trp Thr Gly Arg Phe Glu Ala His
65 70 75 80
Leu Trp Asp Lys His Cys Leu Ala Ala Leu His Asn Lys Lys Lys Gly
85 90 95
Arg Gln Val Tyr Leu Gly Ala Tyr Asp Ser Glu Glu Ala Ala Ala Arg
100 105 110
Ala Tyr Asp Leu Ala Ala Leu Lys Tyr Trp Gly Pro Glu Thr Leu Leu
115 120 125
Asn Phe Pro Val Glu Asp Tyr Ser Ser Glu Met Pro Glu Met Glu Ala
130 135 140
Val Ser Arg Glu Glu Tyr Leu Ala Ser Leu Arg Arg Arg Ser Ser Gly
145 150 155 160
Phe Ser Arg Gly Val Ser Lys Tyr Arg Gly Val Ala Arg His His His
165 170 175
Asn Gly Arg Trp Glu Ala Arg Ile Gly Arg Val Phe Gly Asn Lys Tyr
180 185 190
Leu Tyr Leu Gly Thr Phe Asp Thr Gln Glu Glu Ala Ala Lys Ala Tyr
195 200 205
Asp Leu Ala Ala Ile Glu Tyr Arg Gly Val Asn Ala Val Thr Asn Phe
210 215 220
Asp Ile Ser Cys Tyr Leu Asp His Pro Leu Phe Leu Ala Gln Leu Gln
225 230 235 240
Gln Glu Pro Gln Val Val Pro Ala Leu Asn Gln Glu Pro Gln Pro Asp
245 250 255
Gln Ser Glu Thr Gly Thr Thr Glu Gln Glu Pro Glu Ser Ser Glu Ala
260 265 270
Lys Thr Pro Asp Gly Ser Ala Glu Pro Asp Glu Asn Ala Val Pro Asp
275 280 285
Asp Thr Ala Glu Pro Leu Thr Thr Val Asp Asp Ser Ile Glu Glu Gly
290 295 300
Leu Trp Ser Pro Cys Met Asp Tyr Glu Leu Asp Thr Met Ser Arg Pro
305 310 315 320
Asn Phe Gly Ser Ser Ile Asn Leu Ser Glu Trp Phe Ala Asp Ala Asp
325 330 335
Phe Asp Cys Asn Ile Gly Cys Leu Phe Asp Gly Cys Ser Ala Ala Asp
340 345 350
Glu Gly Ser Lys Asp Gly Val Gly Leu Ala Asp Phe Ser Leu Phe Glu
355 360 365
Ala Gly Asp Val Gln Leu Lys Asp Val Leu Ser Asp Met Glu Glu Gly
370 375 380
Ile Gln Pro Pro Ala Met Ile Ser Val Cys Asn
385 390 395
<210> 201
<211> 430
<212> БЕЛОК
<213> Triadica sebifera
<400> 201
Met Ala Ser Ser Ser Ser Asp Pro Val Leu Lys Ala Glu Leu Gly Ser
1 5 10 15
Ser Gly Gly Gly Cys Ser Ser Gly Gly Gly Gly Glu Ser Ser Glu Ala
20 25 30
Val Ile Ala Asn Asp Gln Leu Leu Leu Tyr Arg Gly Leu Lys Lys Pro
35 40 45
Lys Lys Glu Arg Gly Cys Thr Ala Lys Glu Arg Ile Ser Lys Met Pro
50 55 60
Pro Cys Thr Ala Gly Lys Arg Ser Ser Ile Tyr Arg Gly Val Thr Arg
65 70 75 80
His Arg Trp Thr Gly Arg Tyr Glu Ala His Leu Trp Asp Lys Ser Thr
85 90 95
Trp Asn Gln Asn Gln Asn Lys Lys Gly Lys Gln Val Tyr Leu Gly Ala
100 105 110
Tyr Asp Asp Glu Glu Ala Ala Ala Arg Ala Tyr Asp Leu Ala Ala Leu
115 120 125
Lys Tyr Trp Gly Pro Gly Thr Leu Ile Asn Phe Pro Val Thr Asp Tyr
130 135 140
Thr Arg Asp Leu Glu Glu Met Gln Asn Met Ser Arg Glu Glu Tyr Leu
145 150 155 160
Ala Ser Leu Arg Arg Lys Ser Ser Gly Phe Ser Arg Gly Ile Ser Lys
165 170 175
Tyr Arg Gly Leu Ser Ser Arg Trp Glu Ser Ser Val Gly Arg Met Pro
180 185 190
Gly Ser Glu Tyr Phe Ser Ser Ile Asn Tyr Val Asp Asp Pro Ala Ala
195 200 205
Glu Ser Glu Tyr Val Gly Ser Leu Cys Phe Glu Arg Lys Ile Asp Leu
210 215 220
Thr Ser Tyr Ile Lys Trp Trp Gly Leu Asn Lys Thr Arg Gln Ala Glu
225 230 235 240
Ser Ile Ser Lys Ser Ala Glu Glu Thr Lys Pro Gly Cys Ala Glu Asp
245 250 255
Ile Gly Gly Glu Leu Lys Thr Thr Glu Trp Ala Ile Gln Pro Thr Glu
260 265 270
Pro Tyr Gln Met Pro Arg Leu Gly Met Pro Val His Val Lys Lys His
275 280 285
Lys Gly Ser Lys Ile Ser Ala Leu Ser Val Leu Ser Gln Ser Ala Ala
290 295 300
Phe Lys Ser Leu Gln Glu Lys Ala Ser Lys Lys Gln Glu Asn Ser Thr
305 310 315 320
Asp Asn Asp Glu Asn Glu Asn Lys Asn Thr Asn Thr Asn Lys Ile Asp
325 330 335
Tyr Gly Lys Ala Val Glu Thr Ser Ala Ser His Asp Ser Ser Asn Glu
340 345 350
Arg Pro Val Thr Ala Leu Gly Met Ser Gly Gly Leu Ser Leu Lys Arg
355 360 365
Asn Val Tyr Gln Leu Thr Pro Phe Leu Ser Ala Pro Leu Leu Thr Asn
370 375 380
Tyr Gly Thr Ile Asp Gln Leu Val Asp Pro Ile Leu Trp Ala Ser Leu
385 390 395 400
Val Pro Val Leu Pro Thr Gly Leu Ser Arg Asn Pro Glu Val Thr Lys
405 410 415
Thr Glu Thr Ser Ser Thr Tyr Thr Phe Phe Arg Pro Glu Glu
420 425 430
<210> 202
<211> 1531
<212> ДНК
<213> Solanum tuberosum
<400> 202
ttttaaatca ttgttttatt ttctctttct ttttacaggt ataaaaggtg aaaattgaag
60
caagattgat tgcaagctat gtgtcaccac gttattgata ctttggaaga aatttttact
120
tatatgtctt tgtttaggag taatatttga tatgttttag ttagattttc ttgtcattta
180
tgctttagta taattttagt tatttttatt atatgatcat gggtgaattt tgatacaaat
240
atttttgtca ttaaataaat taatttatca caacttgatt actttcagtg acaaaaaatg
300
tattgtcgta gtaccctttt ttgttgaata tgaataattt tttttatttt gtgacaattg
360
taattgtcac tacttatgat aatatttagt gacatatatg tcgtcggtaa aagcaaacac
420
tttcagtgac aaaataatag atttaatcac aaaattatta acctttttta taataataaa
480
tttatcccta atttatacat ttaaggacaa agtatttttt ttatatataa aaaatagtct
540
ttagtgacga tcgtagtgtt gagtctagaa atcataatgt tgaatctaga aaaatctcat
600
gcagtgtaaa ataaacctca aaaaggacgt tcagtccata gagggggtgt atgtgacacc
660
ccaacctcag caaaagaaaa cctcccttca acaaggacat ttgcggtgct aaacaatttc
720
aagtctcatc acacatatat ttattatata atactaataa agaatagaaa aggaaaggta
780
aacatcatta aatcgtcttt gtatattttt agtgacaact gattgacgaa atctttttcg
840
tcacacaaaa tttttagtga cgaaacatga tttatagatg atgaaattat ttgtccctca
900
taatctaatt tgttgtagtg atcattactc ctttgtttgt tttatttgtc atgttagtcc
960
attaaaaaaa aatatctctc ttcttatgta cgtgaatggt tggaacggat ctattatata
1020
atactaataa agaatagaaa aaggaaagtg agtgaggttc gagggagaga atctgtttaa
1080
tatcagagtc gatcatgtgt caattttatc gatatgaccc taacttcaac tgagtttaac
1140
caattccgat aaggcgagaa atatcatagt attgagtcta gaaaaatctc atgtagtgtg
1200
gggtaaacct cagcaaggac gttgagtcca tagagggggg tgtatgtgac accccaacct
1260
cagcaaaaga aaacctcccc tcaagaagga catttgcggt gctaaacaat ttcaagtctc
1320
atcacacata tatatatatt atataatact aataaataat agaaaaagga aaggtaaaca
1380
tcactaacga cagttgcggt gcaaactgag tgaggtaata aacagcacta acttttattg
1440
gttatgtcaa actcaaagta aaatttctca acttgtttac gtgcctatat ataccatgct
1500
tgttatatgc tcaaagcacc aacaaaattt a
1531
<210> 203
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотидный праймер
<400> 203
cactcgtgct ttccatcatc
20
<210> 204
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотидный праймер
<400> 204
gaaggctgag caacaagagg
20
<210> 205
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотидный праймер
<400> 205
ggcgattttg gattctgc
18
<210> 206
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотидный праймер
<400> 206
cccaaccctt ccgtatacat
20
<210> 207
<211> 1970
<212> ДНК
<213> Zea mays
<400> 207
ggtacctttt ttcccagaga taaatgtgga atagctctac aaacaaacgg catgatgctg
60
acacttggat ggcgaccttg caatcccaag aactattgca tacggttgcc agtcgacaaa
120
tatctacgcc atgcatggct acggtcggaa tacaccgtag cggcgggtaa ctcgccgata
180
ccgtccacgt gtccttggat gcccggtcgc tgatacttct ggtcttctgg acatgcacca
240
agacaatcaa gtgattcaac cttaatttaa cataatataa ataatacgta acatccaact
300
gacgtgttca cctatagaga atattccttc tgattctact ttcagaatga tgccgttgcc
360
gtgtatcgag caagtactct cactcgaagt atcttatctc ccacatccag cacaaaaatc
420
ttctgttcgt ggcaaatctt gtggcggttg aacgaaagaa tgctatataa gtagctatag
480
agaacgtatt atgtgtaaac caaccgttca gtgtaaatcg tgtgtaaata gtcatgttaa
540
ttttttggcg gcagatcaag tacaaactgt atgcctcgga taaacatgta caaaccacaa
600
cactggccac tagatctata tccaacgttc ataaccatcc atccctctct gctgcactct
660
gcaaacaagc acccccatct cgtagcaaca tcttgtctcc gacaagctct cgatgtagtg
720
gaggccctcc accgcaatat cctagtgtat gatgttggag aagcgactcc taaataatgg
780
tggcaagatg ttgctaggtt tgtagccata gcctcaatct aagatcatcc caagccatgg
840
gacctgattc tacgaggcct acaaccaggc atgacacgtc gtctacccac tcttgtgcat
900
catcggtcac ttgatctgac ttggttccta accacttacc ctaggttcca aagccctaag
960
tttctcgtat attgttagtc attcttagtg ggagttttat gtgtatttca ttcctgttaa
1020
atagcatgcc aactaagcaa acatgatgat ataatatgca atctaataaa aagatatatg
1080
agtgggtttc ataaaaaagg gagagagttt catgaggagt gaaactctga atacagatac
1140
tgatatgaca gctttaaaag tagtgttatg aaatcatcat tgagaaatgg tattagcact
1200
caatcgattt ctacgctgtc aattgtcatg agcacaattt tcacccaaag aggcacacca
1260
gcaatgtccg cttgtagtgt ccgagacgtt gctccatcgc cgtcgtcttg tttctgtgcg
1320
ctccattcaa tgcggcaagt ggctcaatcc caagcggtcg tcgcctccca gccccagcag
1380
caaaatatct tcccatgcgg ccatgccttg aaaattggaa tagattctct agattcaccg
1440
ccgcgtcatc ttcactactt tctcactggc ccaatcagca tctccttctc cgagctcaat
1500
catgctcagt caagcgtcac caatggcgtc acggttggtt ttgtcactgt ctgcatgcaa
1560
gggtattttg cttcgcaagt gtaaatggaa aatggatcta aacaactgca ctgcaccaat
1620
tttggaacgc ggaaccgaga gtctgtttgg gttcgtttga aacgcgctga tgtttctcat
1680
tttttaatag atgtagttac ctgatactat ttaagttgga cgatcaaacg actgtgtcaa
1740
gtgtgattaa gaaaagcatc gaaaataaaa tttatcgcca taaaaagtta aaaacagtgg
1800
ataatagtag gacctcataa tagaaaaaat tatcaaacgg aatggagggg cccaacgcag
1860
tatatagcag ccgggtggtg ccggacatcc gacgctcgtg ccagcaggcc attcttctcg
1920
ccttactccc tcacagaacc cagtaaaata tcgccagtcc cgccgtcgag
1970
<210> 208
<211> 584
<212> ДНК
<213> Aeluropus littoralis
<400> 208
cccaagcttg accgatgcac acgctacctg ccaaggctcc ctccatccgc actctgcatc
60
gtcgcttcgg cgtaaacttc cacgtagtac ttgtacgatt ctagctagac ccagtgcgcc
120
caccctaccg ccggcgagcg ggcccccatc tcgcgccagg cttccatgcg ggtccaccgt
180
ggaccagccc tacgccgaac cgagcccatc cctccaccct ttcaccgcca agcgggaccc
240
gcgttggacc tttccgcttg gctggccccc accagcgtcc acgcgggcca acggcctcgc
300
gaaatggatc tccacacgac aaaccaaaac gagaagaaaa taaatggaaa ggaaagaaac
360
ggatcgccac gcgttccaga ggcgtccgct aaccacccga ttatgcttgc gcagcgtgcg
420
taacctcgtc gtggggttaa tccgggtggc cggatcggga aagccacggc ctttataacc
480
catccctgcc ggatcgaacc ggtaccggaa acaaaaacag ggggagaaaa aaagttcttc
540
gcgaggaagg aaaaggaaaa gtcgcgtgcc gtcctcgccc acag
584
<210> 209
<211> 928
<212> ДНК
<213> Agrobacterium rhizogenes
<400> 209
ttagcgaaag gatgtcaaaa aaggatgccc ataattggga ggagtggggt aaagcttaaa
60
gttggcccgc tattggattt cgcgaaagcg gcattggcaa acgtggagat tgctgcattc
120
aagatacttt ttctattttc tggttaagat gtaaagtatt gccacaatca tattaattac
180
taacattgta tatgtaatat agtgcggaaa ttatctatgc caaaatgatg tattaataat
240
agcaataata atatgtgtta atctttttca atcgggaata cgtttaagcg attatcgtgt
300
tgaataaatt attccaaaag gaaatacatg gttttggaga acctgctata gatatatgcc
360
aaatttacac tagtttagtg ggtgcaaaac tattatctct gtttctgagt ttaataaaaa
420
ataaataagc agggcgaata gcagttagcc taagaaggaa tggtggccat gtacgtgctt
480
ttaagagacc ctataataaa ttgccagctg tgttgctttg gtgccgacag gcctaacgtg
540
gggtttagct tgacaaagta gcgcctttcc gcagcataaa taaaggtagg cgggtgcgtc
600
ccattattaa aggaaaaagc aaaagctgag attccataga ccacaaacca ccattattgg
660
aggacagaac ctattccctc acgtgggtcg ctagctttaa acctaataag taaaaacaat
720
taaaagcagg caggtgtccc ttctatattc gcacaacgag gcgacgtgga gcatcgacag
780
ccgcatccat taattaataa atttgtggac ctatacctaa ctcaaatatt tttattattt
840
gctccaatac gctaagagct ctggattata aatagtttgg atgcttcgag ttatgggtac
900
aagcaacctg tttcctactt tgttacca
928
<210> 210
<211> 732
<212> ДНК
<213> Искусственная
<220>
<223> конструкция шпРНКи, содержащая фрагмент 732 пары оснований
<400> 210
aatgcagttt tacaaagtgg agtacccaaa gcagatgaga tcattttgta taacatggct
60
cttgtgttgg atcgtatttt tgtggatgtg aaggatgctt ttgagttctc accacatcat
120
aaggccattc gtgaaccttt tgactattac aagtttggcc aaaattatat ccgcccttta
180
cttgatttca ggagttctta tgttggcaat atatcagttt ttggtgaaat agaagagaag
240
ctcaagcagg gcgttaatgt tgttttgatg tcaaaccacc aaagtgaagc agatccagcg
300
gttattgctc tgttgcttga atcgaggcac ccatacattg ctgagaacat aatttatgtt
360
gcaggagata gagttattac tgatcctctt tgcaagccat tcagcatggg aaggaatctc
420
ctgtgtgttt attcgaaaaa acatatgggt gatgacccca aacttgtcga gaagaaaagg
480
agagcaaaca caagaagctt gaaggagatg gctgtgctat tgaggggtgg atcaaaacta
540
atatggattg ctcctagtgg tggaagagat aggccaaacc ctgttacaaa agaatggtat
600
ccagcgccat ttgatgcttc ttcaacagac aacatgagaa ggcttgtaga acatgctggt
660
gtccctggtc acatttatcc tctagcaata ttatgctatg atattatgcc ccctccgccc
720
caggttgaga aa
732
<210> 211
<211> 512
<212> БЕЛОК
<213> Elaeis guineensis
<400> 211
Met Ala Val Ser Lys Asn Pro Glu Thr Leu Ala Pro Asp Gln Glu Pro
1 5 10 15
Ser Lys Glu Ser Asp Leu Arg Arg Arg Pro Ala Ser Ser Pro Ser Ser
20 25 30
Thr Ala Ala Ser Pro Ala Val Pro Asp Ser Ser Ser Arg Thr Ser Ser
35 40 45
Ser Ile Thr Gly Ser Trp Thr Thr Ala Leu Asp Gly Asp Ser Gly Ala
50 55 60
Gly Ala Val Arg Ile Gly Asp Pro Lys Asp Arg Ile Gly Glu Ala Asn
65 70 75 80
Asp Ile Gly Glu Lys Lys Lys Ala Cys Ser Gly Glu Val Pro Val Gly
85 90 95
Phe Val Asp Arg Pro Ser Ala Pro Val His Val Arg Val Val Glu Ser
100 105 110
Pro Leu Ser Ser Asp Thr Ile Phe Gln Gln Ser His Ala Gly Leu Leu
115 120 125
Asn Leu Cys Val Val Val Leu Ile Ala Val Asn Ser Arg Leu Ile Ile
130 135 140
Glu Asn Leu Met Lys Tyr Gly Leu Leu Ile Gly Ser Gly Phe Phe Phe
145 150 155 160
Ser Ser Arg Leu Leu Arg Asp Trp Pro Leu Leu Ile Cys Ser Leu Thr
165 170 175
Leu Pro Val Phe Pro Leu Gly Ser Tyr Met Val Glu Lys Leu Ala Tyr
180 185 190
Lys Lys Phe Ile Ser Glu Pro Val Val Val Ser Leu His Val Ile Leu
195 200 205
Ile Ile Ala Thr Ile Met Tyr Pro Val Phe Val Ile Leu Arg Cys Asp
210 215 220
Ser Pro Ile Leu Ser Gly Ile Asn Leu Met Leu Phe Val Ser Ser Ile
225 230 235 240
Cys Leu Lys Leu Val Ser Tyr Ala His Ala Asn Tyr Asp Leu Arg Ser
245 250 255
Ser Ser Asn Ser Ile Asp Lys Gly Ile His Lys Ser Gln Gly Val Ser
260 265 270
Phe Lys Ser Leu Val Tyr Phe Ile Met Ala Pro Thr Leu Cys Tyr Gln
275 280 285
Pro Ser Tyr Pro Arg Thr Thr Cys Ile Arg Lys Gly Trp Val Ile Cys
290 295 300
Gln Leu Val Lys Leu Val Ile Phe Thr Gly Val Met Gly Phe Ile Ile
305 310 315 320
Glu Gln Tyr Ile Asp Pro Ile Ile Lys Asn Ser Gln His Pro Leu Lys
325 330 335
Gly Asn Val Leu Asn Ala Met Glu Arg Val Leu Lys Leu Ser Ile Pro
340 345 350
Thr Leu Tyr Val Trp Leu Cys Val Phe Tyr Cys Thr Phe His Leu Trp
355 360 365
Leu Asn Ile Leu Ala Glu Leu Leu Cys Phe Gly Asp Arg Glu Phe Tyr
370 375 380
Lys Asp Trp Trp Asn Ala Lys Thr Ile Glu Glu Tyr Trp Arg Met Trp
385 390 395 400
Asn Met Pro Val His Lys Trp Met Leu Arg His Val Tyr Leu Pro Cys
405 410 415
Ile Arg Asn Gly Ile Pro Lys Gly Val Ala Met Val Ile Ser Phe Phe
420 425 430
Ile Ser Ala Ile Phe His Glu Leu Cys Ile Gly Ile Pro Cys His Ile
435 440 445
Phe Lys Phe Trp Ala Phe Ile Gly Ile Met Phe Gln Val Pro Leu Val
450 455 460
Ile Leu Thr Lys Tyr Leu Gln Asn Lys Phe Lys Ser Ala Met Val Gly
465 470 475 480
Asn Met Ile Phe Trp Phe Phe Phe Ser Ile Tyr Gly Gln Pro Met Cys
485 490 495
Val Leu Leu Tyr Tyr His Asp Val Met Asn Arg Lys Val Gly Thr Glu
500 505 510
<210> 212
<211> 74
<212> БЕЛОК
<213> Glycine max
<400> 212
Met Ala Asp Ile Asp Arg Ser Phe Asp Asn Asn Val Ser Ala Val Ser
1 5 10 15
Thr Glu Lys Ser Ser Gln Val Ser Asp Val Glu Phe Ser Glu Ala Glu
20 25 30
Glu Ile Leu Ile Ala Met Val Tyr Asn Leu Val Gly Glu Arg Trp Ser
35 40 45
Leu Ile Ala Gly Arg Ile Pro Gly Arg Thr Ala Glu Glu Ile Glu Lys
50 55 60
Tyr Trp Thr Ser Arg Phe Ser Thr Ser Gln
65 70
<210> 213
<211> 146
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 213
Met Gly Ser Leu Gln Met Gln Thr Ser Pro Glu Ser Asp Asn Asp Pro
1 5 10 15
Arg Tyr Ala Thr Val Thr Asp Glu Arg Lys Arg Lys Arg Met Ile Ser
20 25 30
Asn Arg Glu Ser Ala Arg Arg Ser Arg Met Arg Lys Gln Lys Gln Leu
35 40 45
Gly Asp Leu Ile Asn Glu Val Thr Leu Leu Lys Asn Asp Asn Ala Lys
50 55 60
Ile Thr Glu Gln Val Asp Glu Ala Ser Lys Lys Tyr Ile Glu Met Glu
65 70 75 80
Ser Lys Asn Asn Val Leu Arg Ala Gln Ala Ser Glu Leu Thr Asp Arg
85 90 95
Leu Arg Ser Leu Asn Ser Val Leu Glu Met Val Glu Glu Ile Ser Gly
100 105 110
Gln Ala Leu Asp Ile Pro Glu Ile Pro Glu Ser Met Gln Asn Pro Trp
115 120 125
Gln Met Pro Cys Pro Met Gln Pro Ile Arg Ala Ser Ala Asp Met Phe
130 135 140
Asp Cys
145
<210> 214
<211> 268
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 214
Met Gly Arg Gly Lys Ile Glu Ile Lys Arg Ile Glu Asn Ala Asn Ser
1 5 10 15
Arg Gln Val Thr Phe Ser Lys Arg Arg Ser Gly Leu Leu Lys Lys Ala
20 25 30
Arg Glu Leu Ser Val Leu Cys Asp Ala Glu Val Ala Val Ile Val Phe
35 40 45
Ser Lys Ser Gly Lys Leu Phe Glu Tyr Ser Ser Thr Gly Met Lys Gln
50 55 60
Thr Leu Ser Arg Tyr Gly Asn His Gln Ser Ser Ser Ala Ser Lys Ala
65 70 75 80
Glu Glu Asp Cys Ala Glu Val Asp Ile Leu Lys Asp Gln Leu Ser Lys
85 90 95
Leu Gln Glu Lys His Leu Gln Leu Gln Gly Lys Gly Leu Asn Pro Leu
100 105 110
Thr Phe Lys Glu Leu Gln Ser Leu Glu Gln Gln Leu Tyr His Ala Leu
115 120 125
Ile Thr Val Arg Glu Arg Lys Glu Arg Leu Leu Thr Asn Gln Leu Glu
130 135 140
Glu Ser Arg Leu Lys Glu Gln Arg Ala Glu Leu Glu Asn Glu Thr Leu
145 150 155 160
Arg Arg Gln Val Gln Glu Leu Arg Ser Phe Leu Pro Ser Phe Thr His
165 170 175
Tyr Val Pro Ser Tyr Ile Lys Cys Phe Ala Ile Asp Pro Lys Asn Ala
180 185 190
Leu Ile Asn His Asp Ser Lys Cys Ser Leu Gln Asn Thr Asp Ser Asp
195 200 205
Thr Thr Leu Gln Leu Gly Leu Pro Gly Glu Ala His Asp Arg Arg Thr
210 215 220
Asn Glu Gly Glu Arg Glu Ser Pro Ser Ser Asp Ser Val Thr Thr Asn
225 230 235 240
Thr Ser Ser Glu Thr Ala Glu Arg Gly Asp Gln Ser Ser Leu Ala Asn
245 250 255
Ser Pro Pro Glu Ala Lys Arg Gln Arg Phe Ser Val
260 265
<210> 215
<211> 437
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 215
Met Glu Phe Glu Ser Val Phe Lys Met His Tyr Pro Tyr Leu Ala Ala
1 5 10 15
Val Ile Tyr Asp Asp Ser Ser Thr Leu Lys Asp Phe His Pro Ser Leu
20 25 30
Thr Asp Asp Phe Ser Cys Val His Asn Val His His Lys Pro Ser Met
35 40 45
Pro His Thr Tyr Glu Ile Pro Ser Lys Glu Thr Ile Arg Gly Ile Thr
50 55 60
Pro Ser Pro Cys Thr Glu Ala Phe Gly Ala Cys Phe His Gly Thr Ser
65 70 75 80
Asn Asp His Val Phe Phe Gly Met Ala Tyr Thr Thr Pro Pro Thr Ile
85 90 95
Glu Pro Asn Val Ser His Val Ser His Asp Asn Thr Met Trp Glu Asn
100 105 110
Asp Gln Asn Gln Gly Phe Ile Phe Gly Thr Glu Ser Thr Leu Asn Gln
115 120 125
Ala Met Ala Asp Ser Asn Gln Phe Asn Met Pro Lys Pro Leu Leu Ser
130 135 140
Ala Asn Glu Asp Thr Ile Met Asn Arg Arg Gln Asn Asn Gln Val Met
145 150 155 160
Ile Lys Thr Glu Gln Ile Lys Lys Lys Asn Lys Arg Phe Gln Met Arg
165 170 175
Arg Ile Cys Lys Pro Thr Lys Lys Ala Ser Ile Ile Lys Gly Gln Trp
180 185 190
Thr Pro Glu Glu Asp Lys Leu Leu Val Gln Leu Val Asp Leu His Gly
195 200 205
Thr Lys Lys Trp Ser Gln Ile Ala Lys Met Leu Gln Gly Arg Val Gly
210 215 220
Lys Gln Cys Arg Glu Arg Trp His Asn His Leu Arg Pro Asp Ile Lys
225 230 235 240
Lys Asp Gly Trp Thr Glu Glu Glu Asp Ile Ile Leu Ile Lys Ala His
245 250 255
Lys Glu Ile Gly Asn Arg Trp Ala Glu Ile Ala Arg Lys Leu Pro Gly
260 265 270
Arg Thr Glu Asn Thr Ile Lys Asn His Trp Asn Ala Thr Lys Arg Arg
275 280 285
Gln His Ser Arg Arg Thr Lys Gly Lys Asp Glu Ile Ser Leu Ser Leu
290 295 300
Gly Ser Asn Thr Leu Gln Asn Tyr Ile Arg Ser Val Thr Tyr Asn Asp
305 310 315 320
Asp Pro Phe Met Thr Ala Asn Ala Asn Ala Asn Ile Gly Pro Arg Asn
325 330 335
Met Arg Gly Lys Gly Lys Asn Val Met Val Ala Val Ser Glu Tyr Asp
340 345 350
Glu Gly Glu Cys Lys Tyr Ile Val Asp Gly Val Asn Asn Leu Gly Leu
355 360 365
Glu Asp Gly Arg Ile Lys Met Pro Ser Leu Ala Ala Met Ser Ala Ser
370 375 380
Gly Ser Ala Ser Thr Ser Gly Ser Ala Ser Gly Ser Gly Ser Gly Val
385 390 395 400
Thr Met Glu Ile Asp Glu Pro Met Thr Asp Ser Trp Met Val Met His
405 410 415
Gly Cys Asp Glu Val Met Met Asn Glu Ile Ala Leu Leu Glu Met Ile
420 425 430
Ala His Gly Arg Leu
435
<210> 216
<211> 359
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 216
Met Tyr His Gln Asn Leu Ile Ser Ser Thr Pro Asn Gln Asn Ser Asn
1 5 10 15
Pro His Asp Trp Asp Ile Gln Asn Pro Leu Phe Ser Ile His Pro Ser
20 25 30
Ala Glu Ile Pro Ser Lys Tyr Pro Phe Met Gly Ile Thr Ser Cys Pro
35 40 45
Asn Thr Asn Val Phe Glu Glu Phe Gln Tyr Lys Ile Thr Asn Asp Gln
50 55 60
Asn Phe Pro Thr Thr Tyr Asn Thr Pro Phe Pro Val Ile Ser Glu Gly
65 70 75 80
Ile Ser Tyr Asn Met His Asp Val Gln Glu Asn Thr Met Cys Gly Tyr
85 90 95
Thr Ala His Asn Gln Gly Leu Ile Ile Gly Cys His Glu Pro Val Leu
100 105 110
Val His Ala Val Val Glu Ser Gln Gln Phe Asn Val Pro Gln Ser Glu
115 120 125
Asp Ile Asn Leu Val Ser Gln Ser Glu Arg Val Thr Glu Asp Lys Val
130 135 140
Met Phe Lys Thr Asp His Lys Lys Lys Asp Ile Ile Gly Lys Gly Gln
145 150 155 160
Trp Thr Pro Thr Glu Asp Glu Leu Leu Val Arg Met Val Lys Ser Lys
165 170 175
Gly Thr Lys Asn Trp Thr Ser Ile Ala Lys Met Phe Gln Gly Arg Val
180 185 190
Gly Lys Gln Cys Arg Glu Arg Trp Arg Asn His Leu Arg Pro Asn Ile
195 200 205
Lys Lys Asn Asp Trp Ser Glu Glu Glu Asp Gln Ile Leu Ile Glu Val
210 215 220
His Lys Ile Val Gly Asn Lys Trp Thr Glu Ile Ala Lys Arg Leu Pro
225 230 235 240
Gly Arg Ser Glu Asn Ile Val Lys Asn His Trp Asn Ala Thr Lys Arg
245 250 255
Arg Leu His Ser Val Arg Thr Lys Arg Ser Asp Ala Phe Ser Pro Arg
260 265 270
Asn Asn Ala Leu Glu Asn Tyr Ile Arg Ser Ile Thr Ile Asn Asn Asn
275 280 285
Ala Leu Met Asn Arg Glu Val Asp Ser Ile Thr Ala Asn Ser Glu Ile
290 295 300
Asp Ser Thr Arg Cys Glu Asn Ile Val Asp Glu Val Met Asn Leu Asn
305 310 315 320
Leu His Ala Thr Thr Ser Val Tyr Val Pro Glu Gln Ala Val Leu Thr
325 330 335
Trp Gly Tyr Asp Phe Thr Lys Cys Tyr Glu Pro Met Asp Asp Thr Trp
340 345 350
Met Leu Met Asn Gly Trp Asn
355
<210> 217
<211> 386
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 217
Met Ser Lys Arg Pro Pro Pro Asp Pro Val Ala Val Leu Arg Gly His
1 5 10 15
Arg His Ser Val Met Asp Val Ser Phe His Pro Ser Lys Ser Leu Leu
20 25 30
Phe Thr Gly Ser Ala Asp Gly Glu Leu Arg Ile Trp Asp Thr Ile Gln
35 40 45
His Arg Ala Val Ser Ser Ala Trp Ala His Ser Arg Ala Asn Gly Val
50 55 60
Leu Ala Val Ala Ala Ser Pro Trp Leu Gly Glu Asp Lys Ile Ile Ser
65 70 75 80
Gln Gly Arg Asp Gly Thr Val Lys Cys Trp Asp Ile Glu Asp Gly Gly
85 90 95
Leu Ser Arg Asp Pro Leu Leu Ile Leu Glu Thr Cys Ala Tyr His Phe
100 105 110
Cys Lys Phe Ser Leu Val Lys Lys Pro Lys Asn Ser Leu Gln Glu Ala
115 120 125
Glu Ser His Ser Arg Gly Cys Asp Glu Gln Asp Gly Gly Asp Thr Cys
130 135 140
Asn Val Gln Ile Ala Asp Asp Ser Glu Arg Ser Glu Glu Asp Ser Gly
145 150 155 160
Leu Leu Gln Asp Lys Asp His Ala Glu Gly Thr Thr Phe Val Ala Val
165 170 175
Val Gly Glu Gln Pro Thr Glu Val Glu Ile Trp Asp Leu Asn Thr Gly
180 185 190
Asp Lys Ile Ile Gln Leu Pro Gln Ser Ser Pro Asp Glu Ser Pro Asn
195 200 205
Ala Ser Thr Lys Gly Arg Gly Met Cys Met Ala Val Gln Leu Phe Cys
210 215 220
Pro Pro Glu Ser Gln Gly Phe Leu His Val Leu Ala Gly Tyr Glu Asp
225 230 235 240
Gly Ser Ile Leu Leu Trp Asp Ile Arg Asn Ala Lys Ile Pro Leu Thr
245 250 255
Ser Val Lys Phe His Ser Glu Pro Val Leu Ser Leu Ser Val Ala Ser
260 265 270
Ser Cys Asp Gly Gly Ile Ser Gly Gly Ala Asp Asp Lys Ile Val Met
275 280 285
Tyr Asn Leu Asn His Ser Thr Gly Ser Cys Thr Ile Arg Lys Glu Ile
290 295 300
Thr Leu Glu Arg Pro Gly Val Ser Gly Thr Ser Ile Arg Val Asp Gly
305 310 315 320
Lys Ile Ala Ala Thr Ala Gly Trp Asp His Arg Ile Arg Val Tyr Asn
325 330 335
Tyr Arg Lys Gly Asn Ala Leu Ala Ile Leu Lys Tyr His Arg Ala Thr
340 345 350
Cys Asn Ala Val Ser Tyr Ser Pro Asp Cys Glu Leu Met Ala Ser Ala
355 360 365
Ser Glu Asp Ala Thr Val Ala Leu Trp Lys Leu Tyr Pro Pro His Lys
370 375 380
Ser Leu
385
<210> 218
<211> 292
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 218
Met Glu Pro Pro Gln His Gln His His His His Gln Ala Asp Gln Glu
1 5 10 15
Ser Gly Asn Asn Asn Asn Asn Lys Ser Gly Ser Gly Gly Tyr Thr Cys
20 25 30
Arg Gln Thr Ser Thr Arg Trp Thr Pro Thr Thr Glu Gln Ile Lys Ile
35 40 45
Leu Lys Glu Leu Tyr Tyr Asn Asn Ala Ile Arg Ser Pro Thr Ala Asp
50 55 60
Gln Ile Gln Lys Ile Thr Ala Arg Leu Arg Gln Phe Gly Lys Ile Glu
65 70 75 80
Gly Lys Asn Val Phe Tyr Trp Phe Gln Asn His Lys Ala Arg Glu Arg
85 90 95
Gln Lys Lys Arg Phe Asn Gly Thr Asn Met Thr Thr Pro Ser Ser Ser
100 105 110
Pro Asn Ser Val Met Met Ala Ala Asn Asp His Tyr His Pro Leu Leu
115 120 125
His His His His Gly Val Pro Met Gln Arg Pro Ala Asn Ser Val Asn
130 135 140
Val Lys Leu Asn Gln Asp His His Leu Tyr His His Asn Lys Pro Tyr
145 150 155 160
Pro Ser Phe Asn Asn Gly Asn Leu Asn His Ala Ser Ser Gly Thr Glu
165 170 175
Cys Gly Val Val Asn Ala Ser Asn Gly Tyr Met Ser Ser His Val Tyr
180 185 190
Gly Ser Met Glu Gln Asp Cys Ser Met Asn Tyr Asn Asn Val Gly Gly
195 200 205
Gly Trp Ala Asn Met Asp His His Tyr Ser Ser Ala Pro Tyr Asn Phe
210 215 220
Phe Asp Arg Ala Lys Pro Leu Phe Gly Leu Glu Gly His Gln Glu Glu
225 230 235 240
Glu Glu Cys Gly Gly Asp Ala Tyr Leu Glu His Arg Arg Thr Leu Pro
245 250 255
Leu Phe Pro Met His Gly Glu Asp His Ile Asn Gly Gly Ser Gly Ala
260 265 270
Ile Trp Lys Tyr Gly Gln Ser Glu Val Arg Pro Cys Ala Ser Leu Glu
275 280 285
Leu Arg Leu Asn
290
<210> 219
<211> 453
<212> БЕЛОК
<213> Brassica napus
<400> 219
Met Asp Leu Gly Ser Val Thr Gly Asn Val Asn Gly Ser Pro Ser Leu
1 5 10 15
Lys Glu Leu Arg Glu Ser Lys Gln Asp Arg Ser Glu Phe Asp Gly Glu
20 25 30
Asp Cys Leu Gln Gln Ser Ser Lys Leu Ala Arg Thr Ile Ala Glu Asp
35 40 45
Lys His Leu Pro Ser Ser Tyr Ala Ala Ala Tyr Ser Arg Pro Met Ser
50 55 60
Phe His Gln Gly Ile Pro Leu Ala Arg Ser Ala Ser Leu Leu Ser Ser
65 70 75 80
Asp Ser Arg Arg Gln Glu His Met Leu Ser Phe Ser Asp Lys Pro Glu
85 90 95
Ala Phe Asp Phe Ser Lys Tyr Val Gly Leu Asp Asn Asn Lys Asn Ser
100 105 110
Leu Ser Pro Phe Leu His Gln Leu Pro Pro Pro Tyr Cys Arg Thr Pro
115 120 125
Gly Gly Gly Tyr Gly Ser Gly Gly Met Met Met Ser Met Gln Gly Lys
130 135 140
Gly Pro Phe Thr Leu Thr Gln Trp Ala Glu Leu Glu Gln Gln Ala Leu
145 150 155 160
Ile Tyr Lys Tyr Ile Thr Ala Asn Val Pro Val Pro Ser Ser Leu Leu
165 170 175
Ile Ser Ile Gln Lys Ser Phe Tyr Pro Tyr Arg Ser Phe Pro Pro Ser
180 185 190
Ser Phe Gly Trp Gly Thr Phe His Leu Gly Phe Ala Gly Gly Lys Met
195 200 205
Asp Pro Glu Pro Gly Arg Cys Arg Arg Thr Asp Gly Lys Lys Trp Arg
210 215 220
Cys Ser Lys Asp Ala Val Pro Asp Gln Lys Tyr Cys Glu Arg His Ile
225 230 235 240
Asn Arg Gly Arg His Arg Ser Arg Lys Pro Val Glu Val Gln Pro Gly
245 250 255
Gln Thr Ala Ala Ser Lys Ala Ala Ala Val Ala Ser Arg Asn Thr Ala
260 265 270
Ser Gln Ile Pro Asn Asn Arg Val Gln Asn Val Ile Tyr Pro Ser Thr
275 280 285
Val Asn Leu Pro Pro Lys Glu Gln Arg Asn Asn Asn Asn Ser Ser Phe
290 295 300
Gly Phe Gly His Val Thr Ser Pro Ser Leu Leu Thr Ser Ser Tyr Leu
305 310 315 320
Asp Phe Ser Ser Asn Gln Asn Lys Pro Glu Glu Leu Lys Ser Asp Trp
325 330 335
Thr Gln Leu Ser Met Ser Ile Pro Val Ala Ser Ser Ser Pro Ser Ser
340 345 350
Thr Ala Gln Asp Lys Thr Thr Leu Ser Pro Leu Arg Leu Asp Leu Pro
355 360 365
Ile Gln Ser Gln Gln Glu Thr Leu Glu Ala Val Arg Lys Val Asn Thr
370 375 380
Trp Ile Pro Ile Ser Trp Gly Asn Ser Leu Gly Gly Pro Leu Gly Glu
385 390 395 400
Val Leu Asn Ser Thr Thr Ser Ser Pro Thr Leu Gly Ser Ser Pro Thr
405 410 415
Gly Val Leu Gln Lys Ser Thr Phe Cys Ser Leu Ser Asn Ser Ser Ser
420 425 430
Val Thr Ser Pro Val Ala Asp Asn Asn Arg Asn Asn Asn Val Asp Tyr
435 440 445
Phe His Tyr Thr Thr
450
<210> 220
<211> 461
<212> БЕЛОК
<213> Brassica napus
<400> 220
Met Asp Leu Gly Ser Val Thr Gly Asn Val Asn Gly Ser Pro Gly Leu
1 5 10 15
Lys Glu Leu Arg Gly Ser Lys Gln Asp Arg Ser Gly Phe Asp Gly Glu
20 25 30
Asp Cys Leu Gln Gln Ser Ser Lys Leu Ala Arg Thr Ile Ala Glu Asp
35 40 45
Lys His Leu Pro Ser Ser Tyr Ala Ala Tyr Ser Arg Pro Met Ser Phe
50 55 60
His Gln Gly Ile Pro Leu Thr Arg Ser Ala Ser Leu Leu Ser Ser Asp
65 70 75 80
Ser Arg Arg Gln Glu His Met Leu Ser Phe Ser Asp Lys Pro Glu Ala
85 90 95
Phe Asp Phe Ser Lys Tyr Val Gly Leu Asp Asn Asn Lys Asn Ser Leu
100 105 110
Ser Pro Phe Leu His Gln Leu Pro Pro Pro Tyr Cys Arg Ser Ser Gly
115 120 125
Gly Gly Tyr Gly Ser Gly Gly Met Met Met Ser Met Gln Gly Lys Gly
130 135 140
Pro Phe Thr Leu Thr Gln Trp Ala Glu Leu Glu Gln Gln Ala Leu Ile
145 150 155 160
Tyr Lys Tyr Ile Thr Ala Asn Val Pro Val Pro Ser Ser Leu Leu Ile
165 170 175
Ser Ile Gln Lys Ser Phe Tyr Pro Tyr Arg Ser Phe Pro Pro Ser Ser
180 185 190
Phe Gly Trp Gly Thr Phe His Leu Gly Phe Ala Gly Gly Lys Met Asp
195 200 205
Pro Glu Pro Gly Arg Cys Arg Arg Thr Asp Gly Lys Lys Trp Arg Cys
210 215 220
Ser Lys Asp Ala Val Pro Glu Gln Lys Tyr Cys Glu Arg His Ile Asn
225 230 235 240
Arg Gly Arg His Arg Ser Arg Lys Pro Val Glu Val Gln Pro Gly Gln
245 250 255
Thr Ala Ala Ser Lys Ala Val Ala Ser Arg Asp Thr Ala Ser Gln Ile
260 265 270
Pro Ser Asn Arg Val Gln Asn Val Ile Tyr Pro Ser Asn Val Asn Leu
275 280 285
Gln Pro Lys Glu Gln Arg Asn Asn Asp Asn Ser Pro Phe Gly Phe Gly
290 295 300
His Val Thr Ser Ser Ser Leu Leu Thr Ser Ser Tyr Leu Asp Phe Ser
305 310 315 320
Ser Asn Gln Glu Lys Pro Ser Gly Asn His His Asn Gln Ser Ser Trp
325 330 335
Pro Glu Glu Leu Lys Ser Asp Trp Thr Gln Leu Ser Met Ser Ile Pro
340 345 350
Val Ala Ser Ser Ser Pro Ser Ser Thr Ala Gln Asp Lys Thr Ala Leu
355 360 365
Ser Pro Leu Arg Leu Asp Leu Pro Ile Gln Ser Gln Gln Glu Thr Leu
370 375 380
Glu Ser Ala Arg Lys Val Asn Thr Trp Ile Pro Ile Ser Trp Gly Asn
385 390 395 400
Ser Leu Gly Gly Pro Leu Gly Glu Val Leu Asn Ser Thr Thr Ser Ser
405 410 415
Pro Thr Leu Gly Ser Ser Pro Thr Gly Val Leu Gln Lys Ser Thr Phe
420 425 430
Cys Ser Leu Ser Asn Ser Ser Ser Val Thr Ser Pro Ile Ala Asp Asn
435 440 445
Asn Arg Asn Asn Asn Val Asp Tyr Phe His Tyr Thr Thr
450 455 460
<210> 221
<211> 409
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 221
Met Glu Ala Arg Pro Val His Arg Ser Gly Ser Arg Asp Leu Thr Arg
1 5 10 15
Thr Ser Ser Ile Pro Ser Thr Gln Lys Pro Ser Pro Val Glu Asp Ser
20 25 30
Phe Met Arg Ser Asp Asn Asn Ser Gln Leu Met Ser Arg Pro Leu Gly
35 40 45
Gln Thr Tyr His Leu Leu Ser Ser Ser Asn Gly Gly Ala Val Gly His
50 55 60
Ile Cys Ser Ser Ser Ser Ser Gly Phe Ala Thr Asn Leu His Tyr Ser
65 70 75 80
Thr Met Val Ser His Glu Lys Gln Gln His Tyr Thr Gly Ser Ser Ser
85 90 95
Asn Asn Ala Val Gln Thr Pro Ser Asn Asn Asp Ser Ala Trp Cys His
100 105 110
Asp Ser Leu Pro Gly Gly Phe Leu Asp Phe His Glu Thr Asn Pro Ala
115 120 125
Ile Gln Asn Asn Cys Gln Ile Glu Asp Gly Gly Ile Ala Ala Ala Phe
130 135 140
Asp Asp Ile Gln Lys Arg Ser Asp Trp His Glu Trp Ala Asp His Leu
145 150 155 160
Ile Thr Asp Asp Asp Pro Leu Met Ser Thr Asn Trp Asn Asp Leu Leu
165 170 175
Leu Glu Thr Asn Ser Asn Ser Asp Ser Lys Asp Gln Lys Thr Leu Gln
180 185 190
Ile Pro Gln Pro Gln Ile Val Gln Gln Gln Pro Ser Pro Ser Val Glu
195 200 205
Leu Arg Pro Val Ser Thr Thr Ser Ser Asn Ser Asn Asn Gly Thr Gly
210 215 220
Lys Ala Arg Met Arg Trp Thr Pro Glu Leu His Glu Ala Phe Val Glu
225 230 235 240
Ala Val Asn Ser Leu Gly Gly Ser Glu Arg Ala Thr Pro Lys Gly Val
245 250 255
Leu Lys Ile Met Lys Val Glu Gly Leu Thr Ile Tyr His Val Lys Ser
260 265 270
His Leu Gln Lys Tyr Arg Thr Ala Arg Tyr Arg Pro Glu Pro Ser Glu
275 280 285
Thr Gly Ser Pro Glu Arg Lys Leu Thr Pro Leu Glu His Ile Thr Ser
290 295 300
Leu Asp Leu Lys Gly Gly Ile Gly Ile Thr Glu Ala Leu Arg Leu Gln
305 310 315 320
Met Glu Val Gln Lys Gln Leu His Glu Gln Leu Glu Ile Gln Arg Asn
325 330 335
Leu Gln Leu Arg Ile Glu Glu Gln Gly Lys Tyr Leu Gln Met Met Phe
340 345 350
Glu Lys Gln Asn Ser Gly Leu Thr Lys Gly Thr Ala Ser Thr Ser Asp
355 360 365
Ser Ala Ala Lys Ser Glu Gln Glu Asp Lys Lys Thr Ala Asp Ser Lys
370 375 380
Glu Val Pro Glu Glu Glu Thr Arg Lys Cys Glu Glu Leu Glu Ser Pro
385 390 395 400
Gln Pro Lys Arg Pro Lys Ile Asp Asn
405
<210> 222
<211> 685
<212> ДНК
<213> Nicotiana benthamiana
<400> 222
aaagtccact ggaagaatac tcttcaacag ttggaaagag ttggacctaa gtcggttggt
60
gtctgtctgt taacagcagc ttttgttggc atggccttca ctatccaatt tgttagagaa
120
ttcactagat tagggttaaa tagatctgtt ggtggggtgt tggcccttgc cttttcaaga
180
gagctaagtc cagttgtcac atcaattgta gttgctgggc gtatcggtag tgcatttgct
240
gcggaactgg gcactatgca ggtatctgag cagactgaca cgttgagagt tcttggtgca
300
aatcctgttg attatttggt gacaccaaga gtgattgctt cttgcgttgc attaccattt
360
ttaaccctaa tgtgctttac agttggaatg gcatccagcg cccttttggc agatggtgtt
420
tatggaatta gcataaacat aatcttagat tctgctctga gagctcttag atcatgggac
480
cttattagtg caatgattaa atcaggggtg tttggtgcta ttatatccat cataagctgt
540
gcttgggggg tcaccacgct gggaggtgcc aaaggggttg gagagtcgac tacttcagca
600
gtagttttat ctcttgttgg catattcata gctgactttg ctctctcttg ctgtttcttc
660
cagggtgctg gcgattccct gaaga
685
<210> 223
<211> 824
<212> БЕЛОК
<213> Solanum tuberosum
<400> 223
Met Asp Ile Ser Asn Glu Ala Lys Val Glu Phe Ile Ser Ile Gly Pro
1 5 10 15
Ser Ser Ile Val Gly Arg Thr Ile Ala Phe Arg Val Leu Phe Cys Lys
20 25 30
Ser Ile Ser Arg Leu Arg His Asn Ile Phe His Phe Leu Ile Tyr Tyr
35 40 45
Leu Tyr Lys Ile Lys Asn Cys Leu Ser Tyr Tyr Leu Thr Pro Leu Ile
50 55 60
Lys Trp Phe His Pro Arg Asn Pro Gln Gly Ile Leu Ala Leu Val Thr
65 70 75 80
Leu Leu Ala Phe Leu Leu Arg Arg Tyr Thr Asn Val Lys Ile Arg Ala
85 90 95
Asp Met Val Tyr Lys Arg Lys Phe Trp Arg Asn Met Met Lys Ser Ala
100 105 110
Leu Thr Tyr Glu Glu Trp Ala His Ala Ala Lys Met Leu Glu Lys Glu
115 120 125
Thr Pro Lys Met Asn Glu Ala Glu Phe Tyr Asp Glu Glu Leu Val Val
130 135 140
Asn Lys Leu Gln Glu Leu Gln His Arg Arg Asn Glu Gly Ser Leu Arg
145 150 155 160
Asp Ile Met Phe Phe Met Arg Ala Asp Leu Val Arg Asn Leu Gly Asn
165 170 175
Met Cys Asn Pro Gln Leu His Lys Gly Arg Leu His Val Pro Lys Leu
180 185 190
Ile Lys Glu Tyr Ile Asp Glu Val Ser Thr Gln Leu Lys Met Val Cys
195 200 205
Asp Tyr Asp Ser Asp Glu Ile Leu Leu Glu Glu Lys Leu Ala Phe Met
210 215 220
His Glu Thr Arg His Ala Phe Gly Arg Thr Ala Leu Leu Leu Ser Gly
225 230 235 240
Gly Ala Ser Leu Gly Ala Phe His Val Gly Val Val Lys Thr Leu Val
245 250 255
Glu His Lys Leu Met Pro Arg Ile Ile Ala Gly Ser Ser Val Gly Ser
260 265 270
Ile Met Cys Ser Val Val Ala Thr Arg Ser Trp Pro Glu Leu Gln Ser
275 280 285
Phe Phe Glu Asn Phe Trp His Val Leu Gln Pro Phe Glu Gln Met Gly
290 295 300
Gly Ile Leu Thr Val Phe Arg Arg Ile Met Arg Gln Gly Ala Val His
305 310 315 320
Glu Ile Arg Gln Leu Gln Val Met Leu Arg His Leu Thr Asn Asn Leu
325 330 335
Thr Phe Gln Glu Ala Tyr Asp Met Thr Gly Arg Val Leu Gly Ile Thr
340 345 350
Val Cys Ser Pro Arg Lys His Glu Pro Pro Arg Cys Leu Asn Tyr Leu
355 360 365
Thr Ser Pro His Val Val Ile Trp Ser Ala Val Thr Ala Ser Cys Ala
370 375 380
Phe Pro Gly Leu Phe Glu Ala Gln Glu Leu Met Ala Lys Asp Arg Ser
385 390 395 400
Gly Asn Leu Val Pro Tyr His Pro Pro Phe His Leu Glu Pro Asp Gln
405 410 415
Ala Ala Ala Ser Gly Ser Ser Ala Arg Arg Trp Arg Asp Gly Ser Leu
420 425 430
Glu Ile Asp Leu Pro Met Met Gln Leu Lys Glu Leu Phe Asn Val Asn
435 440 445
His Phe Ile Val Ser Gln Ala Asn Pro His Ile Ala Pro Leu Leu Arg
450 455 460
Ile Lys Glu Phe Val Arg Ala Tyr Gly Gly Asn Phe Ala Ala Lys Leu
465 470 475 480
Ala His Leu Thr Glu Met Glu Val Lys His Arg Cys Asn Gln Val Leu
485 490 495
Glu Leu Gly Phe Pro Leu Arg Gly Leu Ala Lys Leu Phe Ala Gln Asp
500 505 510
Trp Glu Gly Asp Val Thr Val Val Met Pro Ala Thr Leu Ala Gln Tyr
515 520 525
Leu Lys Ile Ile Gln Asn Pro Ser Thr Leu Glu Val Gln Lys Ala Ala
530 535 540
Asn Gln Gly Arg Arg Cys Thr Trp Glu Lys Leu Ser Ala Ile Lys Ala
545 550 555 560
Asn Cys Gly Ile Glu Leu Ala Leu Asp Glu Cys Val Ala Ile Leu Asn
565 570 575
His Met Arg Arg Leu Lys Arg Ser Ala Glu Arg Ala Ala Ala Ala Ser
580 585 590
Gln Gly Met Ser Ser Ser Thr Val Lys Leu Asn Ala Ser Arg Arg Ile
595 600 605
Pro Ser Trp Asn Cys Ile Ala Arg Glu Asn Ser Thr Gly Ser Leu Glu
610 615 620
Glu Asp Phe His Ala Asp Ala Ser Ser Ser Leu His His His Asn Ala
625 630 635 640
Gly Arg Asn Trp Arg Cys Asn Asn Lys Asn Ala Ala His Asp His His
645 650 655
Gly Ser Asp Ser Glu Ser Glu Asn Ala Asp Asn Asn Ser Trp Thr Arg
660 665 670
Ser Gly Gly Pro Leu Met Arg Thr Thr Ser Ala Asp Lys Phe Ile Asp
675 680 685
Tyr Val Gln Asn Leu Glu Met His Pro Ser Gln Arg Ser Ser Arg Gly
690 695 700
Leu Ser Ile Asp Leu Asn Asn Val Val Val Arg Glu Pro Leu Ser Pro
705 710 715 720
Ser Pro Arg Val Thr Thr Pro Ala Arg Arg Ser Asp Thr Glu Phe Asp
725 730 735
Gln Arg Asp Ile Arg Ile Ile Val Ala Glu Gly Asp Leu Leu Gln Thr
740 745 750
Glu Arg Thr Asn Asn Gly Ile Val Phe Asn Val Val Arg Arg Gly Asp
755 760 765
Leu Thr Pro Ser Asn Arg Ser Leu Asp Ser Glu Asn Asn Ser Cys Phe
770 775 780
His Asp Pro Val Ala Glu Cys Val Gln Leu Glu Asn Pro Glu Lys Asp
785 790 795 800
Met Asp Ile Ser Ser Ala Ser Glu Asp Gly Glu Asn Ala Val Leu Asp
805 810 815
Glu Val Thr Lys Asn Gln Ile Ile
820
<210> 224
<211> 2475
<212> ДНК
<213> Solanum tuberosum
<400> 224
atggatataa gtaatgaggc taaagtagag ttcatttcca taggaccttc ttcaattgta
60
ggtcgaacaa tagcctttcg agttttgttt tgcaaatcaa tatcgcggtt gaggcacaac
120
atttttcatt tcttgatata ttacttgtac aagatcaaga attgtctgtc atactacttg
180
acacctttga tcaaatggtt tcacccgcgt aatccacagg ggatattagc attagtaaca
240
cttctagcct tcttgttgag gcgatatacg aatgtaaaaa tcagggctga tatggtttat
300
aagaggaaat tttggaggaa tatgatgaaa tctgcattaa cttatgagga atgggctcat
360
gctgcgaaaa tgttggagaa agagacacct aaaatgaatg aagcagagtt ttatgatgaa
420
gagttagttg taaataaact tcaagaactt caacatcgtc gtaatgaagg atctttaaga
480
gatattatgt tctttatgag agctgatctt gtgagaaatc tgggtaatat gtgtaatcca
540
cagcttcata agggtaggct tcatgtgcct aaacttatta aggagtatat tgatgaggtt
600
tcaactcagt tgaaaatggt atgtgattat gattcagatg agattttgtt ggaggagaag
660
cttgctttta tgcatgaaac aagacatgct tttggtagga cagcattgct tttaagcggg
720
ggcgcgtctt tgggagcttt tcatgttggt gtggttaaga cattggttga gcacaagctt
780
atgccaagga taattgctgg ttcgagtgtt ggatcgatta tgtgttctgt agttgcaact
840
cggtcttggc ctgagctgca gagttttttt gagaattttt ggcatgtgtt gcagccgttt
900
gaacagatgg gtggaattct aactgttttc aggaggatca tgagacaagg ggctgtacat
960
gagattaggc agttgcaggt gatgttacgc catctcacga ataatcttac tttccaagaa
1020
gcttacgata tgactggtcg agttctaggg attactgttt gctcccctag aaaacatgaa
1080
cctcctagat gtttgaacta cttgacttca cctcatgttg ttatatggag tgctgtgact
1140
gcttcttgcg cgtttcctgg tctgtttgaa gctcaagaac tgatggcaaa ggatagaagt
1200
ggtaatcttg ttccttatca tccaccattt catttggaac ctgatcaggc tgcagcttct
1260
ggttcatctg ctcgtcgatg gagggatggt agcttggaga tcgatctacc tatgatgcag
1320
ctaaaagagc tattcaacgt aaaccacttt atcgtgagcc aggcgaatcc acatattgct
1380
cctttactca ggatcaaaga gtttgtaaga gcttatggag gcaactttgc tgccaagctt
1440
gctcatctta ctgagatgga agtgaagcac agatgcaatc aggtactgga acttggtttt
1500
cccttgaggg gattagccaa gctatttgct caagattggg aaggcgatgt caccgttgta
1560
atgccagcca ctcttgctca gtacttgaag atcatacaga atccctctac tttggaggtt
1620
caaaaagcag caaatcaagg gaggagatgc acttgggaga aactatcagc cattaaggca
1680
aattgtggaa ttgagcttgc tcttgatgag tgtgtagcaa tactcaacca tatgcgtaga
1740
ctaaaaagga gcgcggagag agcagctgct gcttcacaag gcatgtcaag cagcacagtc
1800
aaactcaatg cttctagacg tattccttct tggaattgca ttgcaagaga gaactcaaca
1860
ggctcccttg aagaagactt tcacgcggat gcttcttcct ctcttcatca tcacaatgct
1920
ggtcgaaact ggcgttgtaa taacaagaat gctgcacatg atcatcatgg tagtgacagt
1980
gagtctgaaa acgcggataa taattcttgg acaagatcag gtggtccatt gatgaggaca
2040
acatcagctg ataagtttat tgactatgta caaaacttgg aaatgcatcc ttcacaacga
2100
tcgagcagag gactgagtat tgacctcaac aatgttgtag tcagggagcc tctttctccg
2160
agtccacgag tgacaacacc tgctaggaga tcagatacag aatttgatca aagagacatc
2220
agaattatcg tcgctgaagg tgatttacta cagactgaaa ggactaacaa tgggattgta
2280
ttcaatgtgg taaggagagg agacttaact ccatcaaaca ggagtcttga ttcagaaaac
2340
aacagttgct ttcatgatcc agtggccgaa tgcgtgcaac tcgaaaatcc tgagaaggat
2400
atggatataa gttcagcatc agaagatgga gaaaatgcag tactagatga agtaacaaaa
2460
aatcagatca tataa
2475
<210> 225
<211> 521
<212> БЕЛОК
<213> Solanum tuberosum
<400> 225
Met Ala Ala Ser Ile Gly Ala Leu Lys Ser Ser Pro Ser Ser Asn Asn
1 5 10 15
Cys Ile Asn Glu Arg Arg Asn Asp Ser Thr Arg Ala Val Ser Ser Arg
20 25 30
Asn Leu Ser Phe Ser Ser Ser His Leu Ala Gly Asp Lys Leu Met Pro
35 40 45
Ile Ser Ser Leu Arg Ser Gln Gly Val Arg Phe Asn Val Arg Arg Ser
50 55 60
Ser Leu Ile Val Pro Pro Lys Ala Val Ser Asp Ser Gln Asn Ser Gln
65 70 75 80
Thr Cys Leu Asp Pro Asp Ala Ser Arg Ser Val Leu Gly Ile Ile Leu
85 90 95
Gly Gly Gly Ala Gly Thr Arg Leu Tyr Pro Leu Thr Lys Lys Arg Ala
100 105 110
Lys Pro Ala Val Pro Leu Gly Ala Asn Tyr Arg Leu Ile Asp Ile Pro
115 120 125
Val Ser Asn Cys Leu Asn Ser Asn Ile Ser Lys Ile Tyr Val Leu Thr
130 135 140
Gln Phe Asn Ser Ala Ser Leu Asn Arg His Leu Ser Arg Ala Tyr Ala
145 150 155 160
Ser Asn Met Gly Gly Tyr Lys Asn Glu Gly Phe Val Glu Val Leu Ala
165 170 175
Ala Gln Gln Ser Pro Glu Asn Pro Asp Trp Phe Gln Gly Thr Ala Asp
180 185 190
Ala Val Arg Gln Tyr Leu Trp Leu Phe Glu Glu His Thr Val Leu Glu
195 200 205
Tyr Leu Ile Leu Ala Gly Asp His Leu Tyr Arg Met Asp Tyr Glu Lys
210 215 220
Phe Ile Gln Ala His Arg Glu Thr Asp Ala Asp Ile Thr Val Ala Ala
225 230 235 240
Leu Pro Met Asp Glu Lys Arg Ala Thr Ala Phe Gly Leu Met Lys Ile
245 250 255
Asp Glu Glu Gly Arg Ile Ile Glu Phe Ala Glu Lys Pro Gln Gly Glu
260 265 270
Gln Leu Gln Ala Met Lys Val Asp Thr Thr Ile Leu Gly Leu Asp Asp
275 280 285
Lys Arg Ala Lys Glu Met Pro Phe Ile Ala Ser Met Gly Ile Tyr Val
290 295 300
Ile Ser Lys Asp Val Met Leu Ser Leu Leu Arg Asp Lys Phe Pro Gly
305 310 315 320
Ala Asn Asp Phe Gly Ser Glu Val Ile Pro Gly Ala Thr Ser Leu Gly
325 330 335
Met Arg Val Gln Ala Tyr Leu Tyr Asp Gly Tyr Trp Glu Asp Ile Gly
340 345 350
Thr Ile Glu Ala Phe Tyr Asn Ala Asn Leu Gly Ile Thr Lys Lys Pro
355 360 365
Val Pro Asp Phe Ser Phe Tyr Asp Arg Ser Ala Pro Ile Tyr Thr Gln
370 375 380
Pro Arg Tyr Leu Pro Pro Ser Lys Met Leu Asp Ala Asp Val Thr Asp
385 390 395 400
Ser Val Ile Gly Glu Gly Cys Val Ile Lys Ser Cys Lys Ile His His
405 410 415
Ser Val Val Gly Leu Arg Ser Cys Ile Ser Glu Gly Ala Ile Ile Glu
420 425 430
Asp Ser Leu Leu Met Gly Ala Asp Tyr Tyr Glu Thr Asp Ala Asp Arg
435 440 445
Lys Leu Leu Ala Ala Lys Gly Ser Val Pro Ile Gly Ile Gly Lys Asn
450 455 460
Cys His Ile Lys Arg Ala Ile Ile Asp Lys Asn Ala Arg Ile Gly Asp
465 470 475 480
Asn Val Lys Ile Ile Asn Lys Asp Asn Val Gln Glu Ala Ala Arg Glu
485 490 495
Thr Asp Gly Tyr Phe Ile Lys Ser Gly Ile Val Thr Val Ile Lys Asp
500 505 510
Ala Leu Ile Pro Ser Gly Ile Ile Ile
515 520
<210> 226
<211> 1819
<212> ДНК
<213> Solanum tuberosum
<400> 226
ctagtgattg caatcacact ctaccacaca ctctctagta gagagatcag ttgataacaa
60
gctttgttaa caatggcggc ttccattgga gccttaaaat cttcaccttc ttctaacaat
120
tgcatcaatg agagaagaaa tgattctaca cgtgcagtat ccagcagaaa tctctcattt
180
tcgtcttctc atctcgccgg agacaagttg atgcctatat cgtccttacg ttcccaagga
240
gtccgattca atgtgagaag aagttcattg attgtgccgc ctaaggctgt ttctgattcg
300
cagaattcac agacatgtct agacccagat gctagccgga gtgttttggg aattattctt
360
ggaggtggag ctgggacccg actttatcct ctaactaaaa aaagagcaaa gccagctgtt
420
ccacttggag caaattatcg tctgattgac attcctgtaa gcaactgctt gaacagtaac
480
atatccaaga tctatgttct cacacaattc aactctgcct ctctgaatcg ccacctttca
540
cgagcatatg ctagcaacat gggaggatac aaaaacgagg gctttgtgga agttcttgct
600
gctcaacaaa gtccagagaa ccccgattgg ttccagggca cggctgatgc tgtcagacaa
660
tatctgtggt tgtttgagga gcatactgtt cttgaatacc ttatacttgc tggagatcat
720
ctgtatcgaa tggattatga aaagtttatt caagcccaca gagaaacaga tgctgatatt
780
accgttgccg cactgccaat ggacgagaag cgtgccactg cattcggtct catgaagatt
840
gacgaagaag gacgcattat tgaatttgca gagaaaccgc aaggagagca attgcaagca
900
atgaaagtgg atactaccat tttaggtctt gatgacaaga gagctaaaga aatgcctttc
960
attgccagta tgggtatata tgtcattagc aaagacgtga tgttaagcct acttcgtgac
1020
aagttccctg gggccaatga ttttggtagt gaagttattc ctggtgcaac ttcacttggg
1080
atgagagtgc aagcttattt atatgatggg tactgggaag atattggtac cattgaagct
1140
ttctacaatg ccaatttggg cattacaaaa aagccggtgc cagattttag cttttacgac
1200
cgatcagccc caatctacac ccaacctcga tatctaccac catcaaaaat gcttgatgct
1260
gatgtcacag atagtgtcat tggtgaaggt tgtgtgatca agagctgtaa gattcatcat
1320
tccgtggttg gactcagatc atgcatatca gagggagcaa ttatagaaga ctcacttttg
1380
atgggggcag attactatga gactgatgct gacaggaagt tgctggctgc aaagggcagt
1440
gtcccaattg gcatcggcaa gaattgtcac attaaaagag ccattatcga caagaatgcc
1500
cgtatagggg acaatgtgaa gatcattaac aaagacaacg ttcaagaagc ggctagggaa
1560
acagatggat acttcatcaa gagtgggatt gtcaccgtca tcaaggatgc tttgattcca
1620
agtggaatca tcatctgaag gaatgcgttt taacttggtt gtcctccaag attttggcta
1680
aacagccatg aggtagaaac gtgctgaact tttattttcc tgagctgtag aaatctagtg
1740
tacatctttc tgttatgata cttctcatta cccctacaag agaagactgg atgctgtaaa
1800
aattattcgt ctagaataa
1819
<210> 227
<211> 1173
<212> ДНК
<213> Sapium sebiferum L.
<400> 227
tgccaatagc cagccaataa aacatctaca cgttttcaca cggcttttca tcacagccgt
60
tgtttttctc atctcactcc gtgccttcat cttcatcctc ttctcctctc tctctgtctc
120
tatatgtata gaagcgttag atgtcttgcg ttgttaacca attcattttt cgctttctgc
180
ttcttctaat attataagaa agtttgattc ttcttcttgt caatctttgt tcgcggcttt
240
taacgatatc cgctaaagga aatttgaaat ttcaattatg gccgatggaa acgtcaattc
300
gcaagaacag atggctaagc aggaggaaca gaggctgaag tatttggagt ttgtacaagt
360
ggctgcaata catgctgtgg tgaccttcac aaacctctat gtttatgcca aaaacaagtc
420
gggtccattg aagcccggtg ttgagactgt tgaaggtacg gtcaagagtg tggttggacc
480
tgtttatggc aagttccatg atgttcccat tgaggttctc aagtttgtcg atcgcaagat
540
tgatcaatct gtaagcagcc tagacagccg tgtgcctcca gttgtgaagc agttatcggc
600
ccaagcattt tcagtggctc gcgaagcccc agtggctgct cgtgctgtgg cttctgaagt
660
gcagactgct ggagtgaagg aaactgcatc tgggttggca agaactctgt acttcaaata
720
tgaacccaag gccaaggagc tatacaccaa gtatgaacca aaagcggaac agtgtgctgc
780
ctctgcctgg cgtaagctca atcaactccc agtcttccct catgtagctc aggttgttat
840
gccaacagca gcttattgtt ctgaaaagta caaccaggca gtacttacca ccgctgagaa
900
aggatacaga gtgtcctctt atttgccttt tgtgcccact gagagaattg ctaagttgtt
960
taggaatgag gcacctgaat ctaccccttt cctttccaat tgagcaagat gctgataaat
1020
gattcacaat ggacatgtgg acagaataaa aatctttgga tattatatgg tactgtgtat
1080
ttcaaggttc aagattactc tctacaatgt gtgaattttt gtttcagatg acttaattct
1140
tgttcattca ttatatatat atatatatat ata
1173
<210> 228
<211> 241
<212> БЕЛОК
<213> Sapium sebiferum L.
<400> 228
Met Ala Asp Gly Asn Val Asn Ser Gln Glu Gln Met Ala Lys Gln Glu
1 5 10 15
Glu Gln Arg Leu Lys Tyr Leu Glu Phe Val Gln Val Ala Ala Ile His
20 25 30
Ala Val Val Thr Phe Thr Asn Leu Tyr Val Tyr Ala Lys Asn Lys Ser
35 40 45
Gly Pro Leu Lys Pro Gly Val Glu Thr Val Glu Gly Thr Val Lys Ser
50 55 60
Val Val Gly Pro Val Tyr Gly Lys Phe His Asp Val Pro Ile Glu Val
65 70 75 80
Leu Lys Phe Val Asp Arg Lys Ile Asp Gln Ser Val Ser Ser Leu Asp
85 90 95
Ser Arg Val Pro Pro Val Val Lys Gln Leu Ser Ala Gln Ala Phe Ser
100 105 110
Val Ala Arg Glu Ala Pro Val Ala Ala Arg Ala Val Ala Ser Glu Val
115 120 125
Gln Thr Ala Gly Val Lys Glu Thr Ala Ser Gly Leu Ala Arg Thr Leu
130 135 140
Tyr Phe Lys Tyr Glu Pro Lys Ala Lys Glu Leu Tyr Thr Lys Tyr Glu
145 150 155 160
Pro Lys Ala Glu Gln Cys Ala Ala Ser Ala Trp Arg Lys Leu Asn Gln
165 170 175
Leu Pro Val Phe Pro His Val Ala Gln Val Val Met Pro Thr Ala Ala
180 185 190
Tyr Cys Ser Glu Lys Tyr Asn Gln Ala Val Leu Thr Thr Ala Glu Lys
195 200 205
Gly Tyr Arg Val Ser Ser Tyr Leu Pro Phe Val Pro Thr Glu Arg Ile
210 215 220
Ala Lys Leu Phe Arg Asn Glu Ala Pro Glu Ser Thr Pro Phe Leu Ser
225 230 235 240
Asn
<210> 229
<211> 1252
<212> ДНК
<213> Sapium sebiferum L.
<400> 229
ctacttttcc ctagcattag tattctaggc cccactctgt agattcctcc agctgcctga
60
tctaattttt tatcaactct tgaccgttcg atcatcccaa cggctcagat tcactagtac
120
ttttctcaca ccgtatctcc gattctccat gactccatcg atataaatcg cagtgctcat
180
caactgaatt ctcgaaattg cggttacaag ctgctataag aagcgaaaag aaacgctgag
240
aaacaggatc cgttcctcct ccctcgtttt ttactcctta caagatggag accgagaaga
300
agattcctga attgaagcac ttagggttcg tgaggatggc tgctattcag tcactgattt
360
gcgtctcgaa tctctacgat tacgcgaagc ataactcagg acctttgaga tccactgttg
420
gaaccgtgga gggtgccgta accaccgtag taggtccagt ttaccagaaa ttcaaagacc
480
ttcctgatga tcttcttgta tatgttgata agaaggtgga tgaaggaaca cacaagtttg
540
ataagcatgc tccacctatt gctaagaagg ctgcgagcca agcccatagt ttgtttcata
600
tagccttgga gaaggtcgaa aaactcgtgc aggaggctcg tgcaggagga cctcgtgctg
660
ctctgcattt tgtggctaca gagtcgaagc acttggcgtt gacccaatct gtgaagctgt
720
atagtaaact taatcagttc cctgtcattc acactgttac agatgtaacc cttcccacag
780
ctactcactg gtcagataag tataaccata cccttatgga cctgacccgg aagggttata
840
cgatctttgg ttatttgcct ttggttccta ttgatgacat atctaagaca tttaaacaaa
900
gtaaagcaga ggagaaagaa aatgcaacta cgcataaatc tgattcatcg gattccgact
960
aaacggttgc catcatgtct aatgggtgtg gtttgttaag tatagtggtt tgcgaaaatg
1020
ttctagggtt tatgagcctg ctcgaaagat gctgagaaat ggaaatctgt actatttagg
1080
agtttttccg tactataata atgagtatga atggtttgta aattctgcct tgtgctttct
1140
cgacaagtat atcatgcttc tattttttac tactacttac tggactactg aattgtctca
1200
taattgtccc tagtgtctaa ttaaatatca cctccaaaat attattgaaa aa
1252
<210> 230
<211> 225
<212> БЕЛОК
<213> Sapium sebiferum L.
<400> 230
Met Glu Thr Glu Lys Lys Ile Pro Glu Leu Lys His Leu Gly Phe Val
1 5 10 15
Arg Met Ala Ala Ile Gln Ser Leu Ile Cys Val Ser Asn Leu Tyr Asp
20 25 30
Tyr Ala Lys His Asn Ser Gly Pro Leu Arg Ser Thr Val Gly Thr Val
35 40 45
Glu Gly Ala Val Thr Thr Val Val Gly Pro Val Tyr Gln Lys Phe Lys
50 55 60
Asp Leu Pro Asp Asp Leu Leu Val Tyr Val Asp Lys Lys Val Asp Glu
65 70 75 80
Gly Thr His Lys Phe Asp Lys His Ala Pro Pro Ile Ala Lys Lys Ala
85 90 95
Ala Ser Gln Ala His Ser Leu Phe His Ile Ala Leu Glu Lys Val Glu
100 105 110
Lys Leu Val Gln Glu Ala Arg Ala Gly Gly Pro Arg Ala Ala Leu His
115 120 125
Phe Val Ala Thr Glu Ser Lys His Leu Ala Leu Thr Gln Ser Val Lys
130 135 140
Leu Tyr Ser Lys Leu Asn Gln Phe Pro Val Ile His Thr Val Thr Asp
145 150 155 160
Val Thr Leu Pro Thr Ala Thr His Trp Ser Asp Lys Tyr Asn His Thr
165 170 175
Leu Met Asp Leu Thr Arg Lys Gly Tyr Thr Ile Phe Gly Tyr Leu Pro
180 185 190
Leu Val Pro Ile Asp Asp Ile Ser Lys Thr Phe Lys Gln Ser Lys Ala
195 200 205
Glu Glu Lys Glu Asn Ala Thr Thr His Lys Ser Asp Ser Ser Asp Ser
210 215 220
Asp
225
<210> 231
<211> 938
<212> ДНК
<213> Sapium sebiferum L.
<400> 231
gagtattcac actctggcct gattgggttt gctataaagg gcgatcgttg caacgctcca
60
tattgtctac ttggttttgt ttcaaatctc atcattttgt aaatttgcga cagtgtagcg
120
ttttctagga aaaaggttgc taaaggaaag tagttatcaa accgcagaaa tggcggaatc
180
cgaacttaat caacacacag atatggttca agatgatgat aaaaaactca agtatctaga
240
ttttgtacaa gtggccgcga tctatgttgt ggtttgtttc tctagtatct atgaatatgc
300
taaggaaaac tccggtccac taaaaccagg ggtccaagcc gttgagtgta ccgtcaaaac
360
tgtaataagt ccggtttacg agaagtttcg cgacgtacct tttgaactcc ttaaattcgt
420
cgatcgtaaa gttgacaact ctctaggcga gttggacagg cacgtgccgt cgctggtgaa
480
gcaggcatca agccaagctc gagctgtggc tagtgaaatt caacatgctg gattggtaga
540
cgcaactaag aacattgcga agacgatgta tacaaagtat gaactgacgg cttggcagct
600
ctactgcaaa tacaagccgg tggctaagcg ttacgcggtg tcgacctggc gctcattgaa
660
ccagcttcct ctgtttcctc aagcggctca gattgcaatc ccaactgctg cttcgtggtc
720
tgagaaatac aataagatgg ttcgttacac gaaagataga ggatatccag cggcggtgta
780
tctgccattg atctcggttg agaggattgc caaggtgttc aatgaagact taaacgggcc
840
caccgtccct accaatggat catccgccgc agcacaatag ttttcatttt atgtatttat
900
gtcagattga agacgctccg gagattttga aaacctga
938
<210> 232
<211> 194
<212> БЕЛОК
<213> Sapium sebiferum L.
<400> 232
Met Ala Glu Ser Glu Leu Asn Gln His Thr Asp Met Val Gln Asp Asp
1 5 10 15
Asp Lys Lys Leu Lys Tyr Leu Asp Phe Val Gln Val Ala Ala Ile Tyr
20 25 30
Val Val Val Cys Phe Ser Ser Ile Tyr Glu Tyr Ala Lys Glu Asn Ser
35 40 45
Gly Pro Leu Lys Pro Gly Val Gln Ala Val Glu Cys Thr Val Lys Thr
50 55 60
Val Ile Ser Pro Val Tyr Glu Lys Phe Arg Asp Val Pro Phe Glu Leu
65 70 75 80
Leu Lys Phe Val Asp Arg Lys Val Asp Asn Ser Leu Gly Glu Leu Asp
85 90 95
Arg His Val Pro Ser Leu Val Lys Gln Ala Ser Ser Gln Ala Arg Ala
100 105 110
Val Ala Ser Glu Ile Gln His Ala Gly Leu Val Asp Ala Thr Lys Asn
115 120 125
Ile Ala Lys Thr Met Tyr Thr Lys Tyr Glu Leu Thr Ala Trp Gln Leu
130 135 140
Tyr Cys Lys Tyr Lys Pro Val Ala Lys Arg Tyr Ala Val Ser Thr Trp
145 150 155 160
Arg Ser Leu Asn Gln Leu Pro Leu Phe Pro Gln Ala Ala Gln Ile Ala
165 170 175
Ile Pro Thr Ala Ala Ser Trp Ser Glu Lys Tyr Asn Lys Met Val Arg
180 185 190
Tyr Thr
<210> 233
<211> 2526
<212> ДНК
<213> Sorghum bicolor
<400> 233
atggacgagt ccggggaagc gagcgtcggc tccttcagga tcggcccgtc gacgctgctg
60
ggccgcgggg tggcgctccg cgtgcttctc ttcagctcgc tgtggcgcct gcgggcgcgc
120
gcgtacgccg ccatctcgcg cgtgcgcagc gcggtgctgc cggtggcggc gtcctggctt
180
cacctcagga acacccacgg cgtcctcctc atggtcgtcc tcttcgccct ctccctgagg
240
aagctctccg gcgcgcggtc gcgggcggcg ctcgcgcgcc ggcgcaggca gtacgagaag
300
gccatgctgc atgccgggac gtacgaggtc tgggcccgcg ccgccaatgt gctcgacaag
360
atgtctgatc aggtccatga ggcggatttc tatgacgagg agctgatcag gaacaggctt
420
gaggacctcc ggaggcggag ggaggacggg tcgctgcggg acgtggtgtt ctgtatgcgc
480
ggcgatcttg ttaggaactt ggggaacatg tgcaatcctg aacttcacaa gggcaggcta
540
gaggttccta agcttataaa ggaatacatt gaagaggttt ctattcaact aagaatggtg
600
tgcgaatctg acactgatga gttgctattg ggagagaagc ttgcctttgt tcaggagacc
660
aggcatgcct ttgggaggac agccctactc ttaagtgggg gtgcttcact ggggtctttc
720
catgtaggtg tagtgaaaac attggttgag cataagcttc tgcctcggat tatagcagga
780
tcaagcgttg gttccattat atgttcgatt gttgctaccc ggacatggcc tgagattgag
840
agcttcttca cagactcatt acagaccttg cagttctttg ataggatggg tggaattttt
900
gcagtgatga ggcgagtcac cactcatggt gcactgcatg acattagcca gatgcaaagg
960
cttctgaggg atctcacaag taacttaaca tttcaagagg cttatgacat gactggccgt
1020
gtccttggga tcaccgtttg ctctcctaga aaaaatgagc caccccgctg cctcaactat
1080
ctgacgtcgc cgcacgttgt tatttggagt gctgtaactg cctcttgtgc atttcctggg
1140
ctctttgaag ctcaggaact gatggcgaag gatagattcg gcaacatagt tcccttccat
1200
gcaccctttg ccacagatcc tgaacaaggt cctggagcat caaagcgccg gtggagagat
1260
gggagcctgg aaatggattt gcccatgatg agactcaagg agttgtttaa tgtaaaccat
1320
ttcattgtga gccaaactaa tcctcacatt tctcccctcc tccgaatgaa agagcttgtt
1380
agagtctatg gagggcgctt tgctggaaag cttgctcgtc ttgctgagat ggaggttaag
1440
tatcgatgta accaaatcct agagattggt cttccaatgg gaggacttgc aaaattgttt
1500
gctcaggact gggagggtga tgtcaccatg gttatgccgg caacagtagc tcagtatttg
1560
aagattattc agaatccaac atatgcggag ctccaaatgg ctgccaacca aggccgcagg
1620
tgtacatggg agaagctctc tgcaatcaga gcaaactgtg ccatcgaact tgcattggat
1680
gaatctatag cagttttaaa ccacaaacgg aggctaaaaa gaagcatgga gaggacagag
1740
gctgctttgc agggtcattc taactatgtt cgactcaaaa ctccaaggag ggtaccatca
1800
tggagctgca tcagtcgaga gaattcttca gaatctctct cggaagagat ttcagcagtt
1860
gctacttcaa ccgcgcagca aggtgctgct cttgttgtcg gcacagccac tctttctcac
1920
catgttcgac gcaattctca tgacggaagt gagagtgaat cagaaaccat tgaccttaat
1980
tcctggacca ggagtggtgg gcctctaatg aggacagcat ctgctgacat gttcatcagt
2040
ttcatccata accttgagat tgacacggaa ttaagtaggc cctgtactgt ggagggtggt
2100
actgcaggta tttcgtcaga atctaccttc ccaaatgatc cacaaccgaa caatggctca
2160
agtgttacta ctccaggtag atgcacagaa aattctgaga ccgaggcata cgacactgtc
2220
aacaccagag ccagtcaggc ttctactccc acaagcatcg ctgtttctga aggagatttg
2280
ctgcagcctg aaagcattgc tgacggtatc ctgcttaaca ttgtgaaaag agatgccttg
2340
caggctcaaa atgacagcgt aactgaattg gccgaaagct cctgcactga aacatatgcg
2400
gaaacttgtg acaccatctc agggtctggc actgctgaag ataacaagga tactgctgac
2460
tcaagcaatc actcacttga tattgatgct tttgtagttt cgcatcaacc ttcagctgat
2520
gattag
2526
<210> 234
<211> 3099
<212> ДНК
<213> Triticum aestivum
<400> 234
atgcccgcgc ctgcaggtgc gtgcagccaa gccccaccgc tcgccttcta ttccgcgtcc
60
cctagcttgg cccggccctg ctccgatcca aggccgcggc ggtggcccag tgccctctcc
120
ctcctgccac gccgtccgcc gcccatggac gtcatcacca acgaggcgcg cgtgggggcg
180
ttcgcgatcg gcccgtccac ggcggcgggg cgcgcgctcg cgctgcgcgt gctcctctgc
240
ggctcgctgg cgcggctgcg gcaccgcctc gccgccgcgc tgcgcgccgc ggcgccgctg
300
gcggcggcct ggctgcaccc gcgccacaac acgcggggga tcctgctcgc cgtctgcgcc
360
gtcgcgctgc tgctgcgcgg ccgcgggggc cgcgccgggg tgcgcgcgcg ggtgcagtcc
420
gcctaccgcc gcaagttctg gcggaacatg atgcgcgccg cgctcaccta cgaggagtgg
480
gcgcacgccg cgcggatgct cgagcgggag acgccgcgcc gcgtcaccga cgccgacctc
540
tacgacgagg agctcgtgtg caacaagctc cgtgagctca ggcaccgccg tcaggagggc
600
tcgctcaggg acatcgtctt ctgcatgcgc gccgatctgc tcaggaacct tggtaacatg
660
tgcaaccccg agctccacaa gttgaggctg caggtgccta aaaccatcaa ggagtacatt
720
gaggaggtat ctactcaact gaaaatggtt tgcaattctg attcggacga gttacccctt
780
gaagagaaac tggcatttat gcatgagaca agacatgcct ttggtagatc ggccctactg
840
ctaagtggag gtgcttcatt tggctctttc catgtgggtg ttgtgaaaac cttggtagag
900
cataagcttc tacctaggat tatttcagga tcaagcgttg gcgcaataat gtgtgctatt
960
gtagccacac ggtcatggcc agaactagag agtttttttg aggagtggca ttccttgaaa
1020
ttctttgacc agatgggtgg gatctttcct gtatttaaaa gaattttgac gcatggagcg
1080
gttcatgaca ttaggcactt gcagacgcag ttgagaaatc ttacaagcaa tttgacattt
1140
caagaggcat atgacatgac tggccgggtt ctcgttgtta ctgtgtgttc tccaagaaaa
1200
catgagccac cacgatgcct gaactatttg acatcacctc atgttctcat ttggagtgca
1260
gtaactgctt cctgtgcttt tcctggactt tttgaggccc aggagttgat ggccaaagat
1320
agattcggag aaacagttcc ttttcatgct ccattcttgt tgggtgtgga ggaacgagct
1380
gacgctgcta cacggcgctg gagagatggc agcttagaaa gtgatttacc catgaagcaa
1440
ttgaaggaat tattcaacgt aaatcacttc atagtaagcc aagccaatcc tcacattgct
1500
ccattactga gactaaagga gatcatcagg gcttacggag gcagctttgc tgcaaagctt
1560
gctgaacttg ctgagatgga agttaagcat aggttcaatc aagttctgga acttggattt
1620
ccattaggag gaatagctaa gttgtttgct caacattggg aaggtgatgt gacaatcgtt
1680
atgccagcca cacttgctca gtattcgaag atcatacaga atccttcgta ttctgagctt
1740
cagaaagccg caagtcaggg taggcgatgc acttgggaaa agctctctgc tatcagggca
1800
aactgcgcta ttgagcttgc attagatgaa tgtgttgccc tcctgaacca catgcgtagg
1860
ctgaagagaa gtgcagaaag agcagctgct tcacaaggat atggtgctac aattagactc
1920
tgtccatcta gaaggattcc atcatggaat ctcatagcaa gagaaaattc aactggttct
1980
ctcgatgagg aaatgctcac atgtcccact gttacgagcc atcaagcagt tggagggact
2040
gctgggccat ctaacagaaa tcaccatctc caacatagta tgcatgatag cagtgacagt
2100
gaatctgaga gtatagactt gaactcatgg acgagaagtg gtggccctct catgagaaca
2160
gcctcagcta ataaattcat cagctttgtt cagaaccttg agattgacac agaattcaga
2220
acaatttcac caagggggag cgaaggtgat attgttacac cgaatagtaa cttatttgct
2280
ggtcacccaa ttggtagaga gccagttgat aaccatccag ggcctgctac tcctggtagg
2340
acctcaggca attcaggttg cgatcctcat gatactcctg ttcctaggtc tccatttggt
2400
ctttccacaa gtatcatggt ccctgaaggt gacttgctgc agccggaaaa gattgagaat
2460
ggtattttat tcaatgttgt gagaagggat gctcttgtag cgactactag cggagttgaa
2520
cctcatggat cttcacagga agcagatgtg gaaactgtac cgaccgagtg cctttatggt
2580
gcttcggatg acgacgacga caacgtggaa ctgaatgctg atcatgaagc attatctgac
2640
cctggagatc agagatcctc agttgcagga aacctagatc cgtccacttc catggattgt
2700
caagctgatg aaacaagtac tactcgatca gaagctccat ctctctttaa tatctgtgtg
2760
gagattcctc cagcaaccat gatcagagaa aatagtcggc ccgacgagcc ttcttcagac
2820
ataagactgg agattgtaaa gacagaatgc cctgatgaga attcagctgc tgggaacgat
2880
gaagttggct cagttcctgc caataaagaa tcttcctatt gttctcagac agctgaaaat
2940
agacagcagc atcaagttga tatgggatct gtgaactcct gtagtgtttc agtttcagaa
3000
gatgataggc atgtcagcct catttcgaac gagaaaccag ttactacttc cagtggcgga
3060
gcggagagta tgacatctgg aagaaatgaa gctgactag
3099
<210> 235
<211> 2198
<212> ДНК
<213> Искусственная последовательность
<220>
<223> фрагмент шпРНКи SDP1 S. bicolor
<400> 235
gcggcggcgt ggctgcaccc gcgcgacaac acgcgcggga tcctgctcgc cgtctgcgcc
60
gtcgcgctgg gtgcagtccg cctaccgccg caagttctgg cggaacatga tgcgcgccgc
120
gctcacctac gaggagtggg cgcacgcggc gcggatgctt ggagtgcagt aacagcttcc
180
tgtgcttttc ctggactttt tgaggcccac catctaggag gattccatcc tggaatctca
240
tagcaagaga aaattcaact ggttctctat gtgcaatcct gaacttcaca agggcaggct
300
agaggttcct aagcttataa aggaatacat tgaagaggtt tctattcaac taagaatggt
360
gtgcgaatct gacactgatg agttgctatt gggagagaag cttgcctttg ttcaggagac
420
caggcatgcc tttgggagga cagccctact cttaagtggg ggtgcttcac tggggtcttt
480
ccatgtaggt gtagtgaaaa cattggttga gcataagctt ctgcctcgga ttatagcagg
540
atcaagaagg gtggacccag ctttcttgta caaagtggtc tcgaggaatt cggtacccca
600
gcttggtaag gaaataatta ttttcttttt tccttttagt ataaaatagt taagtgatgt
660
taattagtat gattataata atatagttgt tataattgtg aaaaaataat ttataaatat
720
attgtttaca taaacaacat agtaatgtaa aaaaatatga caagtgatgt gtaagacgaa
780
gaagataaaa gttgagagta agtatattat ttttaatgaa tttgatcgaa catgtaagat
840
gatatactag cattaatatt tgttttaatc ataatagtaa ttctagctgg tttgatgaat
900
taaatatcaa tgataaaata ctatagtaaa aataagaata aataaattaa aataatattt
960
ttttatgatt aatagtttat tatataatta aatatctata ccattactaa atattttagt
1020
ttaaaagtta ataaatattt tgttagaaat tccaatctgc ttgtaattta tcaataaaca
1080
aaatattaaa taacaagcta aagtaacaaa taatatcaaa ctaatagaaa cagtaatcta
1140
atgtaacaaa acataatcta atgctaatat aacaaagcgc aagatctatc attttatata
1200
gtattatttt caatcaacat tcttattaat ttctaaataa tacttgtagt tttattaact
1260
tctaaatgga ttgactatta attaaatgaa ttagtcgaac atgaataaac aaggtaacat
1320
gatagatcat gtcattgtgt tatcattgat cttacatttg gattgattac agttgggaag
1380
ctgggttcga aatcgataag cttgcgctgc agttatcatc atcatcatag acacacgaaa
1440
taaagtaatc agattatcag ttaaagctat gtaatatttg cgccataacc aatcaattaa
1500
aaaatagatc agtttaaaga aagatcaaag ctcaaaaaaa taaaaagaga aaagggtcct
1560
aaccaagaaa atgaaggaga aaaactagaa atttacctgc acaagcttgg atcctctaga
1620
ccactttgta caagaaagct gggtccaccc ttcttgatcc tgctataatc cgaggcagaa
1680
gcttatgctc aaccaatgtt ttcactacac ctacatggaa agaccccagt gaagcacccc
1740
cacttaagag tagggctgtc ctcccaaagg catgcctggt ctcctgaaca aaggcaagct
1800
tctctcccaa tagcaactca tcagtgtcag attcgcacac cattcttagt tgaatagaaa
1860
cctcttcaat gtattccttt ataagcttag gaacctctag cctgcccttg tgaagttcag
1920
gattgcacat agagaaccag ttgaattttc tcttgctatg agattccagg atggaatcct
1980
cctagatggt gggcctcaaa aagtccagga aaagcacagg aagctgttac tgcactccaa
2040
gcatccgcgc cgcgtgcgcc cactcctcgt aggtgagcgc ggcgcgcatc atgttccgcc
2100
agaacttgcg gcggtaggcg gactgcaccc agcgcgacgg cgcagacggc gagcaggatc
2160
ccgcgcgtgt tgtcgcgcgg gtgcagccac gccgccgc
2198
<210> 236
<211> 22
<212> ДНК
<213> Искусственная
<220>
<223> Олигонуклеотидный праймер
<400> 236
ttttaacgat atccgctaaa gg
22
<210> 237
<211> 23
<212> ДНК
<213> Искусственная
<220>
<223> Олигонуклеотидный праймер
<400> 237
aatgaatgaa caagaattaa gtc
23
<210> 238
<211> 22
<212> ДНК
<213> Искусственная
<220>
<223> Олигонуклеотидный праймер
<400> 238
cttttctcac accgtatctc cg
22
<210> 239
<211> 25
<212> ДНК
<213> Искусственная
<220>
<223> Олигонуклеотидный праймер
<400> 239
agcatgatat acttgtcgag aaagc
25
<210> 240
<211> 18
<212> ДНК
<213> Искусственная
<220>
<223> Олигонуклеотидный праймер
<400> 240
gcgacagtgt agcgtttt
18
<210> 241
<211> 25
<212> ДНК
<213> Искусственная
<220>
<223> Олигонуклеотидный праймер
<400> 241
atacataaaa tgaaaactat tgtgc
25
<210> 242
<211> 23
<212> ДНК
<213> Искусственная
<220>
<223> Олигонуклеотидный праймер
<400> 242
acagacatgt ctagacccag atg
23
<210> 243
<211> 24
<212> ДНК
<213> Искусственная
<220>
<223> Олигонуклеотидный праймер
<400> 243
cactctcatc ccaagtgaag ttgc
24
<210> 244
<211> 25
<212> ДНК
<213> Искусственная
<220>
<223> Олигонуклеотидный праймер
<400> 244
ctgagatgga agtgaagcac agatg
25
<210> 245
<211> 21
<212> ДНК
<213> Искусственная
<220>
<223> Олигонуклеотидный праймер
<400> 245
ccattgttag tcctttcagt c
21
<210> 246
<211> 2631
<212> ДНК
<213> Saccharum hybrid
<400> 246
ctgcgacagc tagaggcgcc accgcgtcct agcttcctcc aacttctcgt cggagatccc
60
ttcagggatg cccaatgcca ccgcccctaa gtcaacctgc gggagctgga gcttcgccag
120
ggtcagagct gcggcagcac cctggtagac cgcattcctg atgacccgcg gggtgcgctc
180
catgaagaag tgcattcgcc caaccaagtc gagtgggtcg cctggagggg gcggggaagc
240
aaaacgttgc atgcacctag cgccctggca gcgagctcct gtagtatcac ctgcgtcgcc
300
tccagctcat gctcgcaagc ctccagggcg gcccggcagt gctccaacac tttcgcctcc
360
tcctacagct ccttccacat gcagtcgtgc tccgcacgca ccttctccac ctttttactc
420
ttttctttct cttttcttgg cccatctttg gtattttcac aaatgtcccc ctacaaatga
480
taaatcacca aaactcatgg agcttgctag ttataaactc taattctaag tttggtgttt
540
atttgagtgg attttgtgtg aaagttggtg gttagaaata ggagttaagg accgccaaca
600
agatccccca cacttagccc tttgctcatc ctcgagtaaa gttcaaggac taaggtggaa
660
catctcctca aatggtacga tgcctgcata taagttattc caagcctcac ctatacatgt
720
gaactttgaa gtgtctacca cgccatcttg ggtggttgag aaatggaaca gatcagaatc
780
cagtcatctt tacctctctt gcttagataa cttgggtttt tgtaaggttt tcaaatttaa
840
aacatagtct tgctcctcaa atgattctct catatagctc aatgtgtatg gtttctcacc
900
aaggcaatgt tttgcctctt ttcatcctac ttctaatatt tcttttgtgg agcttagggt
960
agggaatgaa aaggaagcat acttgcattg catatgttac taagtcaaaa accaaatctg
1020
aggagaagca agtcatacaa tctgatcaag atgtgcaagt gtgtggatat gtggattaag
1080
ataactcctg tttattcatg ctctcctcct taataaactt tagagggcat ggcaatcttt
1140
gcatgggcct tcatgagctc atcgtatgtc taagcatgga gctcatcatt tatataagca
1200
tggtgatacc aaaattactc cttttgagca tgtttatatt taggaggacg ttttacctgt
1260
tgaggtaaat ctgaacgcta ataaatcggc taagcaaaat aatttatcac ctgttgattc
1320
taacaatttg atgatggaca atattgatga ggtgactgac aaatgattga aggctttaaa
1380
ggagattgag aaggataaat ctacaataaa aatgtaaaga agaaagcatt caaagtgtga
1440
gatctggtgt ggaagactat tttgcctctt gggggtaaaa gacaacaagt ttagtaagtg
1500
gcctcaaaat tgggagggcc catgcaagat tgttaaagta attgttttgg attgacggag
1560
gcatttcaag gtgatcatct acctagagct ctcaatggga ggtgctcgaa gacatattac
1620
ccatgtgtat ggcaagatgt ttagctagta actgactgat agtgtaaacg atctccaatg
1680
gggcaagaca tattacctaa ggccaggctg gtttttgcaa gttcgagtag gatatagaga
1740
ttctcgtgcg agttgtaaac gatctccaat ggggcaagac atcctaccct atatatagtg
1800
aaggggcagt agctgattga gaatcaatca atcaagcaca atataattta ttaatttttt
1860
attcaaaccc aatttttttc cttttccaac cctaattata gttttccttt tgcctctagg
1920
acaaattgac gtgttccggg tatcctgctg aattaagaac aaccctaggt gcacctgtcc
1980
cgatagagtc ccacctgggt aggcattcat agggattcgt gtatttcctg caaaaaagcg
2040
attaagctgg cttctaaaac tggctaggcc ggattctgtg gccttcacta ccaggtgatt
2100
ttcatgtgat ccgtgcattc tagcactttg ctatgtaacc caaacttaag tcgacaacta
2160
taaatatgct acttgcagga tgttatcacg acacaactcc taatctacgg aagcctaagt
2220
ttagttttgc tcggagacaa gcaattgtgg ccagtcacta tagctacgtc agagggtagt
2280
gggagcagtt gcgtcgttgg attgaaaaca ggtggatcgt atcagatatt atgcattcac
2340
atggacagta aatgtggtac agtaacttcg caaacaataa aatctgtcac aatttattag
2400
tgcactcctc tgacgtaaat gcttctacgt cagaggattt gattccgagg gccgctgcac
2460
ccatcactaa tgacggtctt tacccatcat catggaccat tgttcacatc catgctatca
2520
ctgtcgtcct gtccatgcac tgcagccctc tataaatact ggcatccctc ccccgttcac
2580
agatcacaca acacaagcaa gaaataaacg gtagctgcca taactagtac a
2631
<210> 247
<211> 2907
<212> ДНК
<213> Saccharum hybrid
<400> 247
gcataggcat tgtaaaagcg gtatgcctct tcttcagtgc agaatttcat accaacctta
60
ggtatcctgt cttccataga attttctacc tgagtaggtt cggtctggtt ggatttgtag
120
cgggtttcat gcaaaataag ttagaaatcg tgcaaacttg caatggaggt taaatttgaa
180
atatatttgc atagacaaaa caaatataga ttatgaatgg taatccaata tgacttgcat
240
tttctaactc tattgctact gtgccagatg aagaatgttg atctggagaa gttttgtgag
300
aatgtgacaa caacgggagg tcatatcaag attctgggta cccgcggaga atcggcctcc
360
atgtagttag cctcgtcagg catgggggga attggctgag atgcccccat gtagtcgtca
420
ggcatggaga gtactggctg agatgccatt gttgtgtaga tcgagagaaa cgagaagaat
480
gctagtctaa taataccctt ccgtatgcta accaactatt ataattggca ccatttttca
540
catgctagcg ccttttgcct gctttattta attcaattgg gtccgataag catgtgaacg
600
tgggagacgg ttccgtcgga cggctccgtt ttcttgtagc gtacggcgtg gacggagaaa
660
aggtgagggc ctatctctaa aggggaacga atggatggtg gacacgtgtg gggagacacc
720
gaagggacat gccgaggagg cacacaagct tcagcaggcg tctccagact ctcagaagaa
780
gaagaagctc acggcacggt tgcggctggt tcttgctgtc gctgtctcgt ggtgcacgtt
840
tctgtgatca cgctgaaatc gaccggccgg cggaccaaca ggaggtcagc tcggccactc
900
cgtctccgag cgcatgagtg caccgttcgt ccgcggttcc ttttctcgtg gtgccgtgca
960
cgcctctgcg ttcaccggca ccctgaaacc aatcagaacg ttccctttac aggggaaagg
1020
gacaagtctg ataacctctc tgtttccatc gtcctctaac cgcgaagagc ggacgcacaa
1080
gacttagagt ctatttgttc gaaatttttt actctcacaa aagctagctt ttatagacgg
1140
gcataaaagc tatcatgtcg accggcacgt ttaatattta acttatacca tatgaatatc
1200
atgtcgaact atgaggatga tacttttctg aacgtgattg cgtgagttat taaattgtac
1260
ttttagttgt ttgagcatga aggtctgaac tatgaattta tgatgtattg tggcttgtga
1320
gctactccgc tctacattta gttggtatca taaatattat tatattatca tataaatttg
1380
atcaacttga gatgctttga ctcttcaaga ttcttggaat gacttatcat ttggggtagg
1440
gagtaggttt ctaaggccag tctcagtggg gtttcatcag agtttcatgg acattaaata
1500
agctgatgtg acaccgtatt gatgaagaga gagatgataa gagtttcatg cgagtagaga
1560
gagtttcatg gggatgaaac tcttcttcac tgtttccaaa atatagatgc attggtaaga
1620
gggccatgaa atctctagtg acactgacct aagatgagat tgactctagc actatgtttc
1680
aaaatctgca tgcatgcatg ctttgaatat tgtaacctca cattaactcc cctcacacat
1740
gcatgcaaac gggcggtgca cgcaaaagaa ttgagtgaag atgcacatga aaaataagta
1800
aaatgctttg gcttcatcac ccggcttaaa tgctcgacag aaaaacacgt cggtagtcaa
1860
ggttgtgcct aacaaactgg ggttcacatg taaaacacgt tcatgcctta gaaacggcct
1920
ggagggatta gatacaactt caattatatc ttagggcccc tccaatattg tcagctctaa
1980
actagtttta tgtgtcacgg tggaggagag ggaggctaaa aatataatct tgagctaacg
2040
tgaagagaag agctattttt ttttgctccc caatacatga tagatacaat atgagagaaa
2100
aaatatatga ataaagaaca ctttacatgc cagccataca atatgagatt tcatctaaga
2160
gccaacacct gactcgtact gttgaaggtg tcctagttgg agtggtcgat cttttagttg
2220
ttagtagtgt aagacctagt ttagtgctct tttcttgtct aggtttatgt tgtgttttgg
2280
ctgccaagtg ttgaacaact caaggtaagg tcccatctaa ttctaaaatg atgccaaata
2340
aagatagatt acaaagttaa acgacggaaa aactctaaaa taggatggaa agttttgtag
2400
agtaataatt ggtatgaagt ggcgaagtcg accacaacca aacataaaga gttaaatgca
2460
tggtaggctc ttgatcttgt ctggaggtgc cacttaggtc cacaaactct caaattgcat
2520
ttttgacacc ctaatgttat tcaagtgtgc cacttagatc tacaaactct caaaatgcat
2580
ttctgatacc ctagtgttgt tcaagtgtgt cacttaggca agaaaagtta gataattttg
2640
ataagctatg ggaccaaatt aatttatgta tgcatgctcg aactagttga tgatgatgga
2700
ccccataata gacactagtt catgggctgg tttccttgta tagtactagc tagtataact
2760
ttttcaagtt gtagctacta ctttagctta tactccgcat attacaatca aatagaattc
2820
ggaagtacta taaacgggag cctataaatg gagacgtttt gcatcatgag gctataacaa
2880
cttgagcaaa aacagaagcc gtgcgcc
2907
<210> 248
<211> 1141
<212> ДНК
<213> Saccharum hybrid
<400> 248
actatagggc acgcgtggtc gacggcccgg gctggtctgg ttttggcctc ttttagttac
60
taaattgcca aaaagagtga ctaaaaagtg actaaactga tttagtcctc tagtcaaggg
120
actaaaccag ctaaaagaca tccgctgccc ctcattaatg cacagaagga gagagagagg
180
gagagggagg acattttggt ctttatatag tagctttaat ggactttagt acctagatcc
240
aaaccggtag tgactaaagt ttagtccttg aactgaactt taatccaggg acatggaacc
300
aaacatgccc ttaacttttt tttattctaa tccctcttac attcacttgt ctcacaaagt
360
ggcaagtcat ttgccaccct cactaccagt ggcgactggt taaatatcct catgtttggt
420
tttttttagt aaccaaatac tgcaagctat tgggaaaaaa ggcaaaaaat tatctccttg
480
cttatagttg tataatccat gatccggcaa ttgtttgtta cggagatcct gaatcctctg
540
acgtagagtt taatcaattt tagctcaaga ataatacact ataaagtgga tatgacaatc
600
accgtagtac ttatttatct tgtagtagta tactgtattc gacctgcgat tatgataaag
660
gcatcagaaa ctagagtact ttctagaatc tttagtcagt ttctgtaaga tgaacgtgac
720
taggaaactt atactgttgc aatcctctga cattctctga ttgaaactcg gtttccaaaa
780
atcatatgtt actaaacaaa acatatctaa ccaaatacta tgtggtagtg tagatttata
840
tgctgtgtac tgaaagtgac gtcaagtata gtagtggcag agactcaaaa gatacctgcg
900
gattctgaat accacaacca taaaaaacag gatgatgtta tacttgtccc cttccatgat
960
acaggactgt ttagtaattt cccaaacagc ccataataca ttctgcaccc tttattaaac
1020
ctctactagc tacaacatct tactccatct tgtctagttg gacaagttct ctctttcttg
1080
gctgactcca acttactaca ccgcaacttc ttgtgccctt gttccaacca tcacaattga
1140
g
1141
<210> 249
<211> 4438
<212> ДНК
<213> Saccharum hybrid
<400> 249
aagtacaaac gtagactctg acatacacgc acgtagactc tgacatacac gcataaacga
60
acgaagaatg ttattattta tgttttgagt gggaatattt ggtactgcta tgattcacgt
120
gtgtaaggaa ggattcaaga agaaaggatg cgtttagttc gcgaaaattt ttgactttta
180
ccactatagc actttcgttt gtatttgtta attagtgtcc aatcatggac taattagact
240
caaaagatcc gtctcgtggt tttaaaccaa actgtgtaat taattttttt tatctatatt
300
taatgctcca tatatgtgtc aaatattcga tataacgaag aatcttgaaa atttttagga
360
actaaacatg gccaaagtgt tgtcccgact gagaaacttt ggaagcagaa taaaggctca
420
aaggaacatt taaaagaaga ggatgatata taatcaaaag tggcgacaaa gaagtgtgta
480
cgacccactc gagattgacg aaggacagct tctttgttct tttgtgtgtt actgaatatg
540
taataatctt gtatagattg gtttttaaaa tacggtggca aattaaagac gatatcactt
600
acaaagacat ggacaatgtg gaggggccaa aagttatata aacgacacgc cgaatcggtg
660
atgaacacca catgcctccc ataaagacgg tgtatcaatc tttgatataa tgggtatccg
720
tttgaggcgg catttatact tgatctagtg aaattacaag gagaggaaaa gaagtttaag
780
agaatgataa tgataatgaa aaaaatcgga ggaaaaagaa catgaacaaa gcaagaggag
840
atagccgtgc acacaaaata gagataattt cctcttagaa ctatgaaaac ttcctcttct
900
ttctgcaaca ctgatttgag tttttgttct ctatctagca tttcagtcca tcttgatgtc
960
aagtgacatg taaaaagacg tattgccccc attgctgttt taaattgtct ccacacttga
1020
caacaattta atgagttgtt aaaatattat gtgtgtttat ggccaattat actttttagt
1080
tttgagtttt tcatgaagtc attaagatgc taaaaataat ataaagttgt caatgcttgt
1140
cggaagcccc aatatgtgac taaaatgctg ctaaaagttt atagcatttt tttaaaaatc
1200
taaacaaatt gaaaaaagaa atccaaacta gaaattgtag atcttatcga aaactataag
1260
ttttatataa aaggcgactt tatctaacac cacacaagaa agatgtgctt tttctaagaa
1320
gacaagtctt agtatgtgat taatatgcta ctgaaaattt atattatttt taagcatttt
1380
aataacctca aatggaaaca tacaaaacta agttgcagat cttatcaaga gctataattt
1440
ttatataaaa tgtatattta aataacacca tacaagaaag atatatgatt ttttctaaga
1500
cgacaaagct ttgtatgcaa tttaatatgt tgctaaaaaa tcatattatt ttttttatca
1560
tcttaacgtc ctcaaataaa aaaaaatcag actagttggt atagacctca tcgaggctac
1620
aatttttata aaaactcaac ttcatccggt gttgtataaa aatgatataa tttttcctag
1680
atagagcgtt gccataagtg tattttggtc aagaaatata tgtatactta ttaatgaaat
1740
cctaacaaaa tatactttaa aatctgacgg aaatgttgga taggaaacaa aagcttaaat
1800
caatgctaaa tagggaagtt ttcatcatag ttataatgag tgatttctcc acaaaatatg
1860
atgtaccaca tgttaaatat tactcgcgca caaataatca gagcatatta ctttcatagc
1920
gtggtcgtgg ccatggccta gacttggttg tggacgtctc acttcaccaa ttgatagaaa
1980
aaaaacattt ataagaaaga aaagatacag aaaccatcac acgcgacaac atgacttgcc
2040
gaaacacaaa accaaaaccc aaactcgaga agatgctttc gagaaaaagc ctgaaaagaa
2100
aaaaaatttg cacgtaaaat caaattcgga cggcgaagag ggcaaacgag acagacaact
2160
gggtccactt gctgataaaa aagagagaga ggagggccca cttgccggcg ggcacccctc
2220
agactgtctc caacaatact gacgcaaaca gaagacgcat tggatgcaat gcgttgcgct
2280
gtggcaaaaa attaggtacc tatttctagt gtattccaac agagaacgca aaagaagatg
2340
ccgtactgcg ccatgcattc atgtgggacc ggggaggatg cgggcaacag cagtttgcac
2400
gacccattgg ccggagcatg cgacgtatat ttgcgttgcg cctcgcttcc tacgcaaaat
2460
gtgtcgttgg tatgcctacc ctgttggagg gcgttttctt ctgctaaagt aacgtggagc
2520
acgcatttgc gtaggctgtt ggagatagtc tcaccacgcg gtgaccggac caggccaatt
2580
cccgagccca aaaagaaaaa agcacacaca cagagacaca cgctctcgct ctcgcctccc
2640
tgacgctgga tttaagcaga gcagggagca gaggtgcaac cgcccaccac gatctcccct
2700
cccgcacgcc ccgcgggcag acccagccaa ggcaaggcag ccgcgaaccg gagcacgccg
2760
gccggtgtcg cctcccgcgc cggcggcctg ctgctcgctc gccctcgctt ccgcattgga
2820
tcacgcggcg gttggcgact tggtggtgtc tgctgctggt gattgcgcct agccggccga
2880
cgcggagacg gtgaggcgct gctcttcgct tctctcccca ctgctcccct cagcggtttc
2940
tctctccctg ttatgcgtgg aggagccctg cccccgcgga acggaagcct ccgccggatc
3000
tctgttacgc cgcggttact gcctcgccct ggatttgaac ttgtttcgta attttccctt
3060
gctgcgcttc tcgatttcgg ggaggggttc tgccggcagc tctgccgctc cacctgactt
3120
ggggaccttt ctatgttccg cgacagcagc attgatgatc tgcttgtctc ttgagttttt
3180
ttttcgtgcg atgcatcgag cgcgtgggga cacgatcacg cctgatgggc ggtagtccgc
3240
gatccgcatt tctgaatccc ggcgcctagc cgaggtgcct cggtgcttcc tggttgcctt
3300
gctgctattc ccttcttcgg atccgctctc gtacggctgg cacggtggtt gcggccttag
3360
aatttcgtgg cggcggtttg gttggattgg tgatgctgct ccgtccgcat ttatgaagga
3420
atgttctcca aacttttaag ctgctcgtgt actcggagta ttgaattgcc tgttccttgc
3480
cgctatagga ggccctgggc cagcctaccc cgctttgggt tgtgattggt gatttccggc
3540
agctgttatt gtttcatgat tcgtgtgggg aaaaaaagtt tttttggttc acgagtggtt
3600
tctggtgcat gttttgacaa gttttctatg atgctggtac tgtctttacc cctgctagag
3660
tagtttggtg gtgcgttttc ctattaggtg ggaatttaat cactttccca ctttatcgta
3720
tctctactat ggtaaccatc ttttggcaat tttgattggt atagtcatgt ttaagataag
3780
cttttgaatt caatgatctt gccgttcatt agctagcact taattttgta gagctgcttg
3840
gatcaccaaa gtgccgctca atcttgttca agtgcctatg atatatggga ttctgatgga
3900
actcttagca gtcgtgtcct taggcagtcg gcaccttgat aaggttccaa gagttcaatc
3960
ttacggaaga aatagtgagc ttgatctgag ttcagatcgg ttgtcttcac acttcacgat
4020
taattaccac gtttttaagg tgtgcattct cacttcttta cttccatcgt caatcttctt
4080
aactggttgg gttggaggtg tggtcatgca cccaaccaca taggttgagt cctcttcaac
4140
tcgaatttag gtgcctattt ttttcttaat aaaaaaggcc acctgattct ccttggttgg
4200
tcacattttt ttcttaataa aaaaaggcca cctcaatgtt tctcctttta gcttgagcac
4260
tttttctgga tctcctcttt cttcttaatt ctgatccaag tgtcatcagc gttatattta
4320
tttgaacctg cttgcttttg taagcctgat cagtttgcaa aagttactag aacaatttaa
4380
ccatctgtgc ttgttatttc tgcaggcatc aagtttctaa caatttgaag tacctaaa
4438
<210> 250
<211> 297
<212> ДНК
<213> Sorghum bicolor
<400> 250
atggtggtca gcgcattcac tggacccggg attgggatcg ggttcggtgt cggctgcggg
60
ttcggcgtcg ggtgggggtt cggagggatg cctcttaaca tgtttggctt gggtattggt
120
gggggctgcg gagttggtct tggactagga tggggctttg gaaatgcttt tggttgtcag
180
tatcgatctt caagagtcca gttccagggc attgaatttc agaagaaggt tgaaggagat
240
gaagcaccaa aagttgtttc acaggagctt gctgaaaaat ctcgtcctta tggctag
297
<210> 251
<211> 98
<212> БЕЛОК
<213> Sorghum bicolor
<400> 251
Met Val Val Ser Ala Phe Thr Gly Pro Gly Ile Gly Ile Gly Phe Gly
1 5 10 15
Val Gly Cys Gly Phe Gly Val Gly Trp Gly Phe Gly Gly Met Pro Leu
20 25 30
Asn Met Phe Gly Leu Gly Ile Gly Gly Gly Cys Gly Val Gly Leu Gly
35 40 45
Leu Gly Trp Gly Phe Gly Asn Ala Phe Gly Cys Gln Tyr Arg Ser Ser
50 55 60
Arg Val Gln Phe Gln Gly Ile Glu Phe Gln Lys Lys Val Glu Gly Asp
65 70 75 80
Glu Ala Pro Lys Val Val Ser Gln Glu Leu Ala Glu Lys Ser Arg Pro
85 90 95
Tyr Gly
<210> 252
<211> 297
<212> ДНК
<213> Zea mays
<400> 252
atggtagtca gcacgttcaa tggacccggg attgggatcg ggttcggtgt cggctgcggg
60
ttcggcgtcg ggtgggggtt cggaggaatg cctcttaaca tgttcggctt gggtatcggt
120
gggggttgtg gatttggtct tggactagga tggggctttg gaaatgcttt tggttgtcag
180
tatcgatctt caagagttca gttccaaggc attgaatttc agaagaaggc ggaaggagat
240
gatgcaccaa aagttgtttc accggagctt gctcaaaagt ctcgtcctta tggctag
297
<210> 253
<211> 98
<212> БЕЛОК
<213> Zea mays
<400> 253
Met Val Val Ser Thr Phe Asn Gly Pro Gly Ile Gly Ile Gly Phe Gly
1 5 10 15
Val Gly Cys Gly Phe Gly Val Gly Trp Gly Phe Gly Gly Met Pro Leu
20 25 30
Asn Met Phe Gly Leu Gly Ile Gly Gly Gly Cys Gly Phe Gly Leu Gly
35 40 45
Leu Gly Trp Gly Phe Gly Asn Ala Phe Gly Cys Gln Tyr Arg Ser Ser
50 55 60
Arg Val Gln Phe Gln Gly Ile Glu Phe Gln Lys Lys Ala Glu Gly Asp
65 70 75 80
Asp Ala Pro Lys Val Val Ser Pro Glu Leu Ala Gln Lys Ser Arg Pro
85 90 95
Tyr Gly
<210> 254
<211> 1533
<212> ДНК
<213> Sorghum bicolor
<400> 254
atggcgacgg cgatgggcgc gttggctgcc acctccctga ccccggtccc ggctgccgct
60
acgttccccg gtgatctcgg cctcggacgc cgccgggcgg ctgtgtcagg gtggcgcgcc
120
ggcgggagac ggctgcgtgc gtcgccgcct gcccggaggc cgttcctgtt ctcgccgagg
180
ggcgtttcgg actctcggag ctcgcaaacg tgccttgatc cggacgccag cacgagtgtt
240
cttgggatca tccttggagg tggtgctggg acaaggctgt atccactgac gaagaagagg
300
gcgaaaccag cagtgccgtt gcgcgccaac tacaggctca tagatatccc tgtcagcaac
360
tgtctgaaca gtaacgtctc caagatatat gtgctaacac agttcaactc tgcttcgctc
420
aaccgccacc tctcaagggc ctatgggaac aacattgccg ggtacaagaa tgagggattc
480
gttgaggtcc ttgcagcaca acagagtcca gagaatccca actggtttca gggtactgca
540
gatgctgtgc gtcaatatat gtggctattt gaggagcaca atatcatgga gttccttatt
600
ctggctggag atcacctgta ccgtatggac taccaaaagt tcattcaagc ccatagagaa
660
acagatgctg atataactgt tgcagccctg ccaatggatg aacaacgtgc aactgcattt
720
ggtcttatga aaattgatga tgaagggaga atagttgagt ttgcagaaaa accaaaagga
780
gagaagctga gatcaatgat ggttgacacc actatattgg gccttgatcc tgagagggcc
840
aaggaactgc cttatattgc tagtatggga atctatgttt ttagcaaaga tgtgatgctt
900
cggcttctca gagaaaactt tcctgcagca aatgactttg gaagtgaggt tattcctggc
960
gcaacagaaa ttggattaag ggtgcaagct tacttatatg atggttactg ggaagatatt
1020
ggtactattg aagcatttta taatgcaaac ttgggaataa ccaagaaacc tgtaccggat
1080
tttagcttct atgaccgttc tgctccaatt tatacgcaac ctagatactt gcctccttca
1140
aaggttcttg atgccgatgt gacagacagt gttattggcg aaggttgtgt tattaaacat
1200
tgcacaatca accattctgt agttggactc cgttcctgca tttctgaagg tgcagttata
1260
gaggattctt tgctgatggg tgcagactat tatgagactg aggatgataa gaaagtcctt
1320
tctgagaatg gtggcattcc cattggtatt gggaagaatg cacatatcag aaaagcaata
1380
atcgacaaaa atgctcgtat tggagaaaat gtgaagataa tcaattttga taatgtccaa
1440
gaagcagtaa gggagacgga aggatacttt atcaaaagtg gcattgtcac agtgattaaa
1500
gatgccttaa tccctagtgg aaccatcata taa
1533
<210> 255
<211> 510
<212> БЕЛОК
<213> Sorghum bicolor
<400> 255
Met Ala Thr Ala Met Gly Ala Leu Ala Ala Thr Ser Leu Thr Pro Val
1 5 10 15
Pro Ala Ala Ala Thr Phe Pro Gly Asp Leu Gly Leu Gly Arg Arg Arg
20 25 30
Ala Ala Val Ser Gly Trp Arg Ala Gly Gly Arg Arg Leu Arg Ala Ser
35 40 45
Pro Pro Ala Arg Arg Pro Phe Leu Phe Ser Pro Arg Gly Val Ser Asp
50 55 60
Ser Arg Ser Ser Gln Thr Cys Leu Asp Pro Asp Ala Ser Thr Ser Val
65 70 75 80
Leu Gly Ile Ile Leu Gly Gly Gly Ala Gly Thr Arg Leu Tyr Pro Leu
85 90 95
Thr Lys Lys Arg Ala Lys Pro Ala Val Pro Leu Arg Ala Asn Tyr Arg
100 105 110
Leu Ile Asp Ile Pro Val Ser Asn Cys Leu Asn Ser Asn Val Ser Lys
115 120 125
Ile Tyr Val Leu Thr Gln Phe Asn Ser Ala Ser Leu Asn Arg His Leu
130 135 140
Ser Arg Ala Tyr Gly Asn Asn Ile Ala Gly Tyr Lys Asn Glu Gly Phe
145 150 155 160
Val Glu Val Leu Ala Ala Gln Gln Ser Pro Glu Asn Pro Asn Trp Phe
165 170 175
Gln Gly Thr Ala Asp Ala Val Arg Gln Tyr Met Trp Leu Phe Glu Glu
180 185 190
His Asn Ile Met Glu Phe Leu Ile Leu Ala Gly Asp His Leu Tyr Arg
195 200 205
Met Asp Tyr Gln Lys Phe Ile Gln Ala His Arg Glu Thr Asp Ala Asp
210 215 220
Ile Thr Val Ala Ala Leu Pro Met Asp Glu Gln Arg Ala Thr Ala Phe
225 230 235 240
Gly Leu Met Lys Ile Asp Asp Glu Gly Arg Ile Val Glu Phe Ala Glu
245 250 255
Lys Pro Lys Gly Glu Lys Leu Arg Ser Met Met Val Asp Thr Thr Ile
260 265 270
Leu Gly Leu Asp Pro Glu Arg Ala Lys Glu Leu Pro Tyr Ile Ala Ser
275 280 285
Met Gly Ile Tyr Val Phe Ser Lys Asp Val Met Leu Arg Leu Leu Arg
290 295 300
Glu Asn Phe Pro Ala Ala Asn Asp Phe Gly Ser Glu Val Ile Pro Gly
305 310 315 320
Ala Thr Glu Ile Gly Leu Arg Val Gln Ala Tyr Leu Tyr Asp Gly Tyr
325 330 335
Trp Glu Asp Ile Gly Thr Ile Glu Ala Phe Tyr Asn Ala Asn Leu Gly
340 345 350
Ile Thr Lys Lys Pro Val Pro Asp Phe Ser Phe Tyr Asp Arg Ser Ala
355 360 365
Pro Ile Tyr Thr Gln Pro Arg Tyr Leu Pro Pro Ser Lys Val Leu Asp
370 375 380
Ala Asp Val Thr Asp Ser Val Ile Gly Glu Gly Cys Val Ile Lys His
385 390 395 400
Cys Thr Ile Asn His Ser Val Val Gly Leu Arg Ser Cys Ile Ser Glu
405 410 415
Gly Ala Val Ile Glu Asp Ser Leu Leu Met Gly Ala Asp Tyr Tyr Glu
420 425 430
Thr Glu Asp Asp Lys Lys Val Leu Ser Glu Asn Gly Gly Ile Pro Ile
435 440 445
Gly Ile Gly Lys Asn Ala His Ile Arg Lys Ala Ile Ile Asp Lys Asn
450 455 460
Ala Arg Ile Gly Glu Asn Val Lys Ile Ile Asn Phe Asp Asn Val Gln
465 470 475 480
Glu Ala Val Arg Glu Thr Glu Gly Tyr Phe Ile Lys Ser Gly Ile Val
485 490 495
Thr Val Ile Lys Asp Ala Leu Ile Pro Ser Gly Thr Ile Ile
500 505 510
<210> 256
<211> 1554
<212> ДНК
<213> Zea mays
<400> 256
atggcgatgg cagccatagc ctccccgtcg tcgaggaccc tgatccctcc gcgacaccac
60
ggcgccgcgc cctccccgtc cacctccggc gactcctcgc tccgcctcct ctgcgcacac
120
ccgcgccacg gacggcgcgg ccgggcgatg tccgtctcga cgcccgcggc gcggagccgg
180
ccgttcgtct tctccccgcg cgcggtgtcc gactctaaga gctcccagac ctgcctcgac
240
cccgacgcca gcacgagtgt tcttggaatc attctcggag gtggtgctgg gactagattg
300
taccccttga caaagaagcg tgccaagcct gcagtgccat tgggtgccaa ctatagactg
360
attgatatcc ccgtcagcaa ttgtctcaac agcaacatat ccaagatcta tgtgctaaca
420
caattcaact ctgcttccct caaccgtcac ctctcaagag cctacgggaa caacattgga
480
gggtacaaga atgacgggtt cgttgaagtc ttagctgcac agcagagccc agataatcca
540
aactggtttc agggtactgc agatgctgta aggcaatact tgtggttatt tgaggaacat
600
aatgtgatgg agtttctaat tcttgctggc gatcacctgt accggatgga ttatgaaaag
660
ttcattcagg cacacagaga aacggatgct gatattactg ttgctgccct accaatggat
720
gagaaacgtg caaccgcatt tggcctcatg aaaattgacg aagaagggag gattattgag
780
tttgctgaga aaccgaaagg agatcagttg aaagcaatga tggttgacac caccatactt
840
ggccttgatg acgagagggc aaaggaaatg ccttatattg ctagcatggg tatatatgtt
900
tttagcaagg atgtaatgct tcagctcctc cgtgaacaat ttcctggagc caatgatttt
960
ggaagtgagg ttattccagg tgcaacaagc attggaaaga gggttcaggc ttatctatat
1020
gatggttatt gggaagatat tggtacaatt gaggcatttt ataatgcaaa cttgggaata
1080
accaagaagc caataccaga tttcagcttc tatgaccgtt ctgctccaat ctatacacaa
1140
cctcgacatc tgccaccttc aaaggttctt gatgctgatg tgacagacag tgttattggt
1200
gagggatgtg ttattaaaaa ctgcaagata caccattctg tagttggact ccgttcttgc
1260
atatctgaag gtgctatcat agaggacact ttactaatgg gtgcggacta ctatgagact
1320
gaagctgaca agaaactcct tgccgaaaat ggtggcattc ccattggtat tgggaagaat
1380
tcacacatca gaaaagcaat cattgacaag aatgctcgaa ttggagataa tgtgaagata
1440
ctcaacgctg acaatgttca agaagctgca agggagacag acgggtactt catcaaaggt
1500
ggaattgtca cagtgatcaa ggatgcttta ctccctagtg ggacagttat atga
1554
<210> 257
<211> 517
<212> БЕЛОК
<213> Zea mays
<400> 257
Met Ala Met Ala Ala Ile Ala Ser Pro Ser Ser Arg Thr Leu Ile Pro
1 5 10 15
Pro Arg His His Gly Ala Ala Pro Ser Pro Ser Thr Ser Gly Asp Ser
20 25 30
Ser Leu Arg Leu Leu Cys Ala His Pro Arg His Gly Arg Arg Gly Arg
35 40 45
Ala Met Ser Val Ser Thr Pro Ala Ala Arg Ser Arg Pro Phe Val Phe
50 55 60
Ser Pro Arg Ala Val Ser Asp Ser Lys Ser Ser Gln Thr Cys Leu Asp
65 70 75 80
Pro Asp Ala Ser Thr Ser Val Leu Gly Ile Ile Leu Gly Gly Gly Ala
85 90 95
Gly Thr Arg Leu Tyr Pro Leu Thr Lys Lys Arg Ala Lys Pro Ala Val
100 105 110
Pro Leu Gly Ala Asn Tyr Arg Leu Ile Asp Ile Pro Val Ser Asn Cys
115 120 125
Leu Asn Ser Asn Ile Ser Lys Ile Tyr Val Leu Thr Gln Phe Asn Ser
130 135 140
Ala Ser Leu Asn Arg His Leu Ser Arg Ala Tyr Gly Asn Asn Ile Gly
145 150 155 160
Gly Tyr Lys Asn Asp Gly Phe Val Glu Val Leu Ala Ala Gln Gln Ser
165 170 175
Pro Asp Asn Pro Asn Trp Phe Gln Gly Thr Ala Asp Ala Val Arg Gln
180 185 190
Tyr Leu Trp Leu Phe Glu Glu His Asn Val Met Glu Phe Leu Ile Leu
195 200 205
Ala Gly Asp His Leu Tyr Arg Met Asp Tyr Glu Lys Phe Ile Gln Ala
210 215 220
His Arg Glu Thr Asp Ala Asp Ile Thr Val Ala Ala Leu Pro Met Asp
225 230 235 240
Glu Lys Arg Ala Thr Ala Phe Gly Leu Met Lys Ile Asp Glu Glu Gly
245 250 255
Arg Ile Ile Glu Phe Ala Glu Lys Pro Lys Gly Asp Gln Leu Lys Ala
260 265 270
Met Met Val Asp Thr Thr Ile Leu Gly Leu Asp Asp Glu Arg Ala Lys
275 280 285
Glu Met Pro Tyr Ile Ala Ser Met Gly Ile Tyr Val Phe Ser Lys Asp
290 295 300
Val Met Leu Gln Leu Leu Arg Glu Gln Phe Pro Gly Ala Asn Asp Phe
305 310 315 320
Gly Ser Glu Val Ile Pro Gly Ala Thr Ser Ile Gly Lys Arg Val Gln
325 330 335
Ala Tyr Leu Tyr Asp Gly Tyr Trp Glu Asp Ile Gly Thr Ile Glu Ala
340 345 350
Phe Tyr Asn Ala Asn Leu Gly Ile Thr Lys Lys Pro Ile Pro Asp Phe
355 360 365
Ser Phe Tyr Asp Arg Ser Ala Pro Ile Tyr Thr Gln Pro Arg His Leu
370 375 380
Pro Pro Ser Lys Val Leu Asp Ala Asp Val Thr Asp Ser Val Ile Gly
385 390 395 400
Glu Gly Cys Val Ile Lys Asn Cys Lys Ile His His Ser Val Val Gly
405 410 415
Leu Arg Ser Cys Ile Ser Glu Gly Ala Ile Ile Glu Asp Thr Leu Leu
420 425 430
Met Gly Ala Asp Tyr Tyr Glu Thr Glu Ala Asp Lys Lys Leu Leu Ala
435 440 445
Glu Asn Gly Gly Ile Pro Ile Gly Ile Gly Lys Asn Ser His Ile Arg
450 455 460
Lys Ala Ile Ile Asp Lys Asn Ala Arg Ile Gly Asp Asn Val Lys Ile
465 470 475 480
Leu Asn Ala Asp Asn Val Gln Glu Ala Ala Arg Glu Thr Asp Gly Tyr
485 490 495
Phe Ile Lys Gly Gly Ile Val Thr Val Ile Lys Asp Ala Leu Leu Pro
500 505 510
Ser Gly Thr Val Ile
515
<210> 258
<211> 2049
<212> ДНК
<213> Sorghum bicolor
<400> 258
atgtcgctcc tgcggcggcg gaagcagccg cagccgccgc cgccgccctc ggacggcgac
60
gggtccgacc acgacgacag cgacaagggg aagaagccgt cctcgtcctc gtcctccgcg
120
ccgccgtcca aggaggccac gaggcggacc aaggccaagt ggtcgtgcgt ggacagctgc
180
tgctggctgg tcgggtgcgt gtgctccgcc tggtggctgc tgctctttct ctacaacgcg
240
atgccggcct cgttcccgca gtatgtcacg gaggccatca cggggccgct cccggaccct
300
cccggggtca agctgcagaa ggaggggctc cgtgttaagc accccgtcgt cttcgtcccc
360
ggcatcgtca ccggagggct cgagctatgg gagggccacc agtgcgccga ggggctcttc
420
cgcaagcggc tatggggcgg cacatttggt gacgtataca agagacctct atgctgggtt
480
gaacatatgt ctttggacaa cgaaacagga ttagacaaac ctggaataag ggttaggcca
540
gtcacaggcc ttgttgcagc agactatttc gttcctggat attttgtttg ggctgtctta
600
attgccaatt tagctcgtat tggatatgaa gaaaagacca tgtacatggc tgcatatgat
660
tggaggttat ctttccagaa cactgaggtt cgtgatcaaa ctttgagcag aataaagagc
720
aacattgaac tcatggtagc aacaaatggt ggaaataggg tggtagtgat cccgcactcc
780
atgggggtcc tctattttct gcattttatg aaatgggttg aagcacctcc tcccatggga
840
ggcggcggcg gtccaaactg gtgtgagaag catattaaag ctgtaatgaa tattggtgga
900
cctttcttag gagttcccaa ggcagttgct gggcttttct catctgaagc caaagatgtt
960
gccgttgcta gagctattgc tcctgatgtt ctggactccg attttcttgg gctccaaact
1020
ttgcgccatt tgatgcgtat gacccgaaca tgggattcga caatgtcaat gattcctaaa
1080
ggtggtgata caatttgggg aaatctggat tggtctccag aagatggcct tgaatgtaaa
1140
gctaagaagc acaaaaccaa tgataccgag gtttctaagg atagcaatgg ggaaaatgtc
1200
gaagttcaac ctgagcctat taactatgga aggctggtat cctttggtaa agatgtagcg
1260
gaagcacctt cttcagagat tgagcagata gaatttcgtg atgctgttaa aggtaatagt
1320
attgcccatt caaatacttc atgccgggag atctggacag aatatcatga attaggatgg
1380
ggtggaataa aggcagttga ggactacaaa gtttacactg ctagttctgt tatagacctc
1440
cttcacttcg ttgctccaag gatgatgcag cgtggaaatg tccacttctc atatggaatt
1500
gctgataact tggatgatcc gaaataccaa cattacaaat attggtcaaa ccccttggaa
1560
acaaagctac cgaatgctcc tgacatggaa atattttcga tgtacggagt aggcattcct
1620
accgaaaggg catatgtcta taaggttgcc ccgcaggcag agtgtaatat acctttccgg
1680
attgactcct cggctgaagg tggggaggaa aatagctgct tgaaaggggg tgtttactta
1740
gctgatggtg atgaaactgt tccagttctt agtgcgggct acatgtgtgc aaaaggatgg
1800
cgtggcaaaa ctcgtttcaa ccctgccggc agcaagactt acgtgagaga atacagccat
1860
tcaccaccct cgactctcct ggaaggcagg ggcactcaga gcggtgcaca tgttgatata
1920
atggggaact ttgctctgat tgaggacatc atcagaatag ctgctggggc aaccggtgag
1980
gaaattggtg gcgaccaggt ttattcagat atattcaaat ggtcagagaa gatcaaattg
2040
aaattgtaa
2049
<210> 259
<211> 682
<212> БЕЛОК
<213> Sorghum bicolor
<400> 259
Met Ser Leu Leu Arg Arg Arg Lys Gln Pro Gln Pro Pro Pro Pro Pro
1 5 10 15
Ser Asp Gly Asp Gly Ser Asp His Asp Asp Ser Asp Lys Gly Lys Lys
20 25 30
Pro Ser Ser Ser Ser Ser Ser Ala Pro Pro Ser Lys Glu Ala Thr Arg
35 40 45
Arg Thr Lys Ala Lys Trp Ser Cys Val Asp Ser Cys Cys Trp Leu Val
50 55 60
Gly Cys Val Cys Ser Ala Trp Trp Leu Leu Leu Phe Leu Tyr Asn Ala
65 70 75 80
Met Pro Ala Ser Phe Pro Gln Tyr Val Thr Glu Ala Ile Thr Gly Pro
85 90 95
Leu Pro Asp Pro Pro Gly Val Lys Leu Gln Lys Glu Gly Leu Arg Val
100 105 110
Lys His Pro Val Val Phe Val Pro Gly Ile Val Thr Gly Gly Leu Glu
115 120 125
Leu Trp Glu Gly His Gln Cys Ala Glu Gly Leu Phe Arg Lys Arg Leu
130 135 140
Trp Gly Gly Thr Phe Gly Asp Val Tyr Lys Arg Pro Leu Cys Trp Val
145 150 155 160
Glu His Met Ser Leu Asp Asn Glu Thr Gly Leu Asp Lys Pro Gly Ile
165 170 175
Arg Val Arg Pro Val Thr Gly Leu Val Ala Ala Asp Tyr Phe Val Pro
180 185 190
Gly Tyr Phe Val Trp Ala Val Leu Ile Ala Asn Leu Ala Arg Ile Gly
195 200 205
Tyr Glu Glu Lys Thr Met Tyr Met Ala Ala Tyr Asp Trp Arg Leu Ser
210 215 220
Phe Gln Asn Thr Glu Val Arg Asp Gln Thr Leu Ser Arg Ile Lys Ser
225 230 235 240
Asn Ile Glu Leu Met Val Ala Thr Asn Gly Gly Asn Arg Val Val Val
245 250 255
Ile Pro His Ser Met Gly Val Leu Tyr Phe Leu His Phe Met Lys Trp
260 265 270
Val Glu Ala Pro Pro Pro Met Gly Gly Gly Gly Gly Pro Asn Trp Cys
275 280 285
Glu Lys His Ile Lys Ala Val Met Asn Ile Gly Gly Pro Phe Leu Gly
290 295 300
Val Pro Lys Ala Val Ala Gly Leu Phe Ser Ser Glu Ala Lys Asp Val
305 310 315 320
Ala Val Ala Arg Ala Ile Ala Pro Asp Val Leu Asp Ser Asp Phe Leu
325 330 335
Gly Leu Gln Thr Leu Arg His Leu Met Arg Met Thr Arg Thr Trp Asp
340 345 350
Ser Thr Met Ser Met Ile Pro Lys Gly Gly Asp Thr Ile Trp Gly Asn
355 360 365
Leu Asp Trp Ser Pro Glu Asp Gly Leu Glu Cys Lys Ala Lys Lys His
370 375 380
Lys Thr Asn Asp Thr Glu Val Ser Lys Asp Ser Asn Gly Glu Asn Val
385 390 395 400
Glu Val Gln Pro Glu Pro Ile Asn Tyr Gly Arg Leu Val Ser Phe Gly
405 410 415
Lys Asp Val Ala Glu Ala Pro Ser Ser Glu Ile Glu Gln Ile Glu Phe
420 425 430
Arg Asp Ala Val Lys Gly Asn Ser Ile Ala His Ser Asn Thr Ser Cys
435 440 445
Arg Glu Ile Trp Thr Glu Tyr His Glu Leu Gly Trp Gly Gly Ile Lys
450 455 460
Ala Val Glu Asp Tyr Lys Val Tyr Thr Ala Ser Ser Val Ile Asp Leu
465 470 475 480
Leu His Phe Val Ala Pro Arg Met Met Gln Arg Gly Asn Val His Phe
485 490 495
Ser Tyr Gly Ile Ala Asp Asn Leu Asp Asp Pro Lys Tyr Gln His Tyr
500 505 510
Lys Tyr Trp Ser Asn Pro Leu Glu Thr Lys Leu Pro Asn Ala Pro Asp
515 520 525
Met Glu Ile Phe Ser Met Tyr Gly Val Gly Ile Pro Thr Glu Arg Ala
530 535 540
Tyr Val Tyr Lys Val Ala Pro Gln Ala Glu Cys Asn Ile Pro Phe Arg
545 550 555 560
Ile Asp Ser Ser Ala Glu Gly Gly Glu Glu Asn Ser Cys Leu Lys Gly
565 570 575
Gly Val Tyr Leu Ala Asp Gly Asp Glu Thr Val Pro Val Leu Ser Ala
580 585 590
Gly Tyr Met Cys Ala Lys Gly Trp Arg Gly Lys Thr Arg Phe Asn Pro
595 600 605
Ala Gly Ser Lys Thr Tyr Val Arg Glu Tyr Ser His Ser Pro Pro Ser
610 615 620
Thr Leu Leu Glu Gly Arg Gly Thr Gln Ser Gly Ala His Val Asp Ile
625 630 635 640
Met Gly Asn Phe Ala Leu Ile Glu Asp Ile Ile Arg Ile Ala Ala Gly
645 650 655
Ala Thr Gly Glu Glu Ile Gly Gly Asp Gln Val Tyr Ser Asp Ile Phe
660 665 670
Lys Trp Ser Glu Lys Ile Lys Leu Lys Leu
675 680
<210> 260
<211> 2037
<212> ДНК
<213> Zea mays
<400> 260
atgtcgctcc tgcggcggcg gaagcagcag cagctaccgc cctcggaggg cgacgggtcc
60
gaccacgacg acaacgacaa ggggaagaag ccgtcctcgt cctccgcgcc gccgtccaag
120
gagcccacga ggcggaccaa ggccaagtgg tcgtgcgtgg acagctgctg ctggctggtc
180
gggtgcgtgt gctccgcctg gtggttgctg ctctttctct acaacgcgat gccagcttcg
240
ttcccgcagt atgtcaccga ggccatcacg gggccgctcc cggacccgcc cggggtcaag
300
ctgcagaagg aggggctgcg agctaagcac cccgtcgtct ttgtccccgg catcgtcacc
360
gggggcctcg agctatggga gggacaccaa tgcgctgagg gtctcttccg caagcggcta
420
tggggcggca catttggtga cgtatacaag agacctctat gctgggttga acatatgtcg
480
ttggacaatg aaactggatt agacaaacct ggaataaggg tcaggtcagt cacaggcctt
540
gttgcagcag actatttcgt ccctggatat tttgtttggg ctgtcttaat tgccaattta
600
gctcgtattg gatatgaaga aaagaccatg tacatggctg catatgattg gaggttatct
660
ttccagaaca ctgaggttcg tgatcaaact ttgagcagaa taaagagcaa tattgaactc
720
atggtagcaa caaatggtgg aaatagggtg gtggtgatcc cacactccat gggggtcctc
780
tattttctgc attttatgaa atgggtcgaa gcacctcctc ccatgggggg cggtggtggt
840
ccgaactggt gtgagaagca tattaaagct gtaatgaata ttggaggacc tttcttagga
900
gttcccaagg ctgttgctgg gcttttctca tctgaagcca aagatgttgc cgttgctaga
960
gctatcgctc ctgatgtcct ggactctgat tttcttggac ttcaaacttt gcgccatttg
1020
atgcgtatga cccgaacatg ggattcaaca atgtcaatgc ttcctaaagg tggtgataca
1080
atttggggaa atctggattg gtctccagaa gatggccttg aatgtaaagc taagaagcat
1140
aaaaccaatg ataccgaggt ttctaaggat agcaatgggg aaaatatcga agttcaacct
1200
gaacctataa actacggaag gctggtatcc ttcggtaaag atgtagcaga ggcaccttct
1260
tcagagattg aacagataga atttcgtgat gctgttaaag gtaacgatat cgtccattca
1320
aatgcatcat gccgggagat ctggacagag taccatgaat taggatgggg tggaataaag
1380
gcagtcgcag actacaaagt ttacactgcc agttctgtta tagaccttct tcactttgtt
1440
gctccaagga tgatgcagcg tggaaatgtc cacttttcat atggaattgc tgataacttg
1500
gatgatccga aatatcaaca ttacaaatat tggtcaaacc ccttggaaac aaagctaccg
1560
aatgctcctg acatggaaat aatttccatg tacggagtag gcattcctac tgaaagggca
1620
tatgtctaca agttggctcc acaggcagag tgctatatac cattccggat tgacgcctcg
1680
gctgatggcg gggaggaaaa caaatgcttg aaagggggtg tttacttagc tgacggcgac
1740
gaaactgttc cagttcttag cgcgggctac atgtgtgcaa aagggtggcg tggcaaaact
1800
cgtttcaacc ctgccggcag caagacttac gtgagagagt acagccattc accaccctca
1860
actctcctgg aaggcagggg cactcagagc ggtgcacatg ttgatataat ggggaacttc
1920
gctttgatcg aggacatcat caggatagct gccggggcaa ccggtgagga aattggtggc
1980
gaccaggttt attcagatat attcaaatgg tcagagaaaa tcaaattgaa attgtaa
2037
<210> 261
<211> 678
<212> БЕЛОК
<213> Zea mays
<400> 261
Met Ser Leu Leu Arg Arg Arg Lys Gln Gln Gln Leu Pro Pro Ser Glu
1 5 10 15
Gly Asp Gly Ser Asp His Asp Asp Asn Asp Lys Gly Lys Lys Pro Ser
20 25 30
Ser Ser Ser Ala Pro Pro Ser Lys Glu Pro Thr Arg Arg Thr Lys Ala
35 40 45
Lys Trp Ser Cys Val Asp Ser Cys Cys Trp Leu Val Gly Cys Val Cys
50 55 60
Ser Ala Trp Trp Leu Leu Leu Phe Leu Tyr Asn Ala Met Pro Ala Ser
65 70 75 80
Phe Pro Gln Tyr Val Thr Glu Ala Ile Thr Gly Pro Leu Pro Asp Pro
85 90 95
Pro Gly Val Lys Leu Gln Lys Glu Gly Leu Arg Ala Lys His Pro Val
100 105 110
Val Phe Val Pro Gly Ile Val Thr Gly Gly Leu Glu Leu Trp Glu Gly
115 120 125
His Gln Cys Ala Glu Gly Leu Phe Arg Lys Arg Leu Trp Gly Gly Thr
130 135 140
Phe Gly Asp Val Tyr Lys Arg Pro Leu Cys Trp Val Glu His Met Ser
145 150 155 160
Leu Asp Asn Glu Thr Gly Leu Asp Lys Pro Gly Ile Arg Val Arg Ser
165 170 175
Val Thr Gly Leu Val Ala Ala Asp Tyr Phe Val Pro Gly Tyr Phe Val
180 185 190
Trp Ala Val Leu Ile Ala Asn Leu Ala Arg Ile Gly Tyr Glu Glu Lys
195 200 205
Thr Met Tyr Met Ala Ala Tyr Asp Trp Arg Leu Ser Phe Gln Asn Thr
210 215 220
Glu Val Arg Asp Gln Thr Leu Ser Arg Ile Lys Ser Asn Ile Glu Leu
225 230 235 240
Met Val Ala Thr Asn Gly Gly Asn Arg Val Val Val Ile Pro His Ser
245 250 255
Met Gly Val Leu Tyr Phe Leu His Phe Met Lys Trp Val Glu Ala Pro
260 265 270
Pro Pro Met Gly Gly Gly Gly Gly Pro Asn Trp Cys Glu Lys His Ile
275 280 285
Lys Ala Val Met Asn Ile Gly Gly Pro Phe Leu Gly Val Pro Lys Ala
290 295 300
Val Ala Gly Leu Phe Ser Ser Glu Ala Lys Asp Val Ala Val Ala Arg
305 310 315 320
Ala Ile Ala Pro Asp Val Leu Asp Ser Asp Phe Leu Gly Leu Gln Thr
325 330 335
Leu Arg His Leu Met Arg Met Thr Arg Thr Trp Asp Ser Thr Met Ser
340 345 350
Met Leu Pro Lys Gly Gly Asp Thr Ile Trp Gly Asn Leu Asp Trp Ser
355 360 365
Pro Glu Asp Gly Leu Glu Cys Lys Ala Lys Lys His Lys Thr Asn Asp
370 375 380
Thr Glu Val Ser Lys Asp Ser Asn Gly Glu Asn Ile Glu Val Gln Pro
385 390 395 400
Glu Pro Ile Asn Tyr Gly Arg Leu Val Ser Phe Gly Lys Asp Val Ala
405 410 415
Glu Ala Pro Ser Ser Glu Ile Glu Gln Ile Glu Phe Arg Asp Ala Val
420 425 430
Lys Gly Asn Asp Ile Val His Ser Asn Ala Ser Cys Arg Glu Ile Trp
435 440 445
Thr Glu Tyr His Glu Leu Gly Trp Gly Gly Ile Lys Ala Val Ala Asp
450 455 460
Tyr Lys Val Tyr Thr Ala Ser Ser Val Ile Asp Leu Leu His Phe Val
465 470 475 480
Ala Pro Arg Met Met Gln Arg Gly Asn Val His Phe Ser Tyr Gly Ile
485 490 495
Ala Asp Asn Leu Asp Asp Pro Lys Tyr Gln His Tyr Lys Tyr Trp Ser
500 505 510
Asn Pro Leu Glu Thr Lys Leu Pro Asn Ala Pro Asp Met Glu Ile Ile
515 520 525
Ser Met Tyr Gly Val Gly Ile Pro Thr Glu Arg Ala Tyr Val Tyr Lys
530 535 540
Leu Ala Pro Gln Ala Glu Cys Tyr Ile Pro Phe Arg Ile Asp Ala Ser
545 550 555 560
Ala Asp Gly Gly Glu Glu Asn Lys Cys Leu Lys Gly Gly Val Tyr Leu
565 570 575
Ala Asp Gly Asp Glu Thr Val Pro Val Leu Ser Ala Gly Tyr Met Cys
580 585 590
Ala Lys Gly Trp Arg Gly Lys Thr Arg Phe Asn Pro Ala Gly Ser Lys
595 600 605
Thr Tyr Val Arg Glu Tyr Ser His Ser Pro Pro Ser Thr Leu Leu Glu
610 615 620
Gly Arg Gly Thr Gln Ser Gly Ala His Val Asp Ile Met Gly Asn Phe
625 630 635 640
Ala Leu Ile Glu Asp Ile Ile Arg Ile Ala Ala Gly Ala Thr Gly Glu
645 650 655
Glu Ile Gly Gly Asp Gln Val Tyr Ser Asp Ile Phe Lys Trp Ser Glu
660 665 670
Lys Ile Lys Leu Lys Leu
675
<210> 262
<211> 846
<212> ДНК
<213> Sorghum bicolor
<400> 262
atgccgccgc ccagcctcac cgcggccgcc gccaccacca caacgcgccg ccgcaaggac
60
cacccggcgc cgggcggagg cgcgggggcg aaggagatgg gcgcggcggc ggcgtccgcg
120
gcggaggggt gggcgcggcg gccggagtgg tgctcggcgg cgggcgtggc gggcgtgctg
180
cggcggcacc cggcgcccgc gctcttcggg tgcggcctcc tgctcttcat ggccgtcgag
240
tacaccatcc ccatggtcag gccggactcc ccgccgctcg acctgggatt catcgccacc
300
aggaacatgc acgccgccgt cgccgccacg ccctggctca actcgctcct cgccgcgctc
360
aacacggtca tcgtcgcgat gcaggcggcg tacatcctgt gggcgatcct ggcggagcag
420
cggccgcggg cggccgtcgc ggcgctgatg atgttcacct gccggggcgt gctgggctgc
480
gccacgcagc tgccgctgcc cgaggagttc ctggggtccg ggatggactt ccccgtgggc
540
aacgtctcct tcttcctctt cttctcgggc cacgtcgcgg gcgcggtgat cgcggccgcc
600
gacatgcgcc gcgagggacg ggcggcgctc gcgcgcctct acgacgcgct caacgtgctc
660
caggcggtca ggctgctcgc gtgcagggga cactacacca tcgacctggc tgtcggcgtc
720
ggggccgggg tcctcttcga cacgctctcc gggtggtact tcgacgccaa gaacggcgac
780
ggcaagaacg cgcccgagaa gcactgccgt agctgccagt gccacaaggc tctcctctca
840
cactag
846
<210> 263
<211> 281
<212> БЕЛОК
<213> Sorghum bicolor
<400> 263
Met Pro Pro Pro Ser Leu Thr Ala Ala Ala Ala Thr Thr Thr Thr Arg
1 5 10 15
Arg Arg Lys Asp His Pro Ala Pro Gly Gly Gly Ala Gly Ala Lys Glu
20 25 30
Met Gly Ala Ala Ala Ala Ser Ala Ala Glu Gly Trp Ala Arg Arg Pro
35 40 45
Glu Trp Cys Ser Ala Ala Gly Val Ala Gly Val Leu Arg Arg His Pro
50 55 60
Ala Pro Ala Leu Phe Gly Cys Gly Leu Leu Leu Phe Met Ala Val Glu
65 70 75 80
Tyr Thr Ile Pro Met Val Arg Pro Asp Ser Pro Pro Leu Asp Leu Gly
85 90 95
Phe Ile Ala Thr Arg Asn Met His Ala Ala Val Ala Ala Thr Pro Trp
100 105 110
Leu Asn Ser Leu Leu Ala Ala Leu Asn Thr Val Ile Val Ala Met Gln
115 120 125
Ala Ala Tyr Ile Leu Trp Ala Ile Leu Ala Glu Gln Arg Pro Arg Ala
130 135 140
Ala Val Ala Ala Leu Met Met Phe Thr Cys Arg Gly Val Leu Gly Cys
145 150 155 160
Ala Thr Gln Leu Pro Leu Pro Glu Glu Phe Leu Gly Ser Gly Met Asp
165 170 175
Phe Pro Val Gly Asn Val Ser Phe Phe Leu Phe Phe Ser Gly His Val
180 185 190
Ala Gly Ala Val Ile Ala Ala Ala Asp Met Arg Arg Glu Gly Arg Ala
195 200 205
Ala Leu Ala Arg Leu Tyr Asp Ala Leu Asn Val Leu Gln Ala Val Arg
210 215 220
Leu Leu Ala Cys Arg Gly His Tyr Thr Ile Asp Leu Ala Val Gly Val
225 230 235 240
Gly Ala Gly Val Leu Phe Asp Thr Leu Ser Gly Trp Tyr Phe Asp Ala
245 250 255
Lys Asn Gly Asp Gly Lys Asn Ala Pro Glu Lys His Cys Arg Ser Cys
260 265 270
Gln Cys His Lys Ala Leu Leu Ser His
275 280
<210> 264
<211> 849
<212> ДНК
<213> Zea mays
<400> 264
atgccgccgc ccagcctcac cgccgccggc accaccacca ccacaacccg ccgccgcaac
60
gaccgagccg cgaaggtcca ccaggtactg ggcgaaggcg cggggacgga ggagatgggc
120
gcggtggcgg acgggtggac gcggcccgag tggtgctcgg cggcgggcgt cgcgggcgtg
180
ctgcggcggc acccggcgcc cgcgctcttc gggtgcggcc tcctgctctt catggccgtc
240
gagtacacca tccccatggt caagccggac gcgccgccgc tcgacctagg cttcctcgcc
300
accgcgggca tgcacgccgc catcgccgcg aggccctggc ttaactcgct cctcgccgcg
360
ctcaacacgg tcttcgtcgc gatgcaggcg gcgtacatcc tgtgggccat cctcgccgag
420
cagcggccgc gcgcggccgt cgccgcgctc atgatgttca cttgccgggg cgtgctgggc
480
tgcgccaccc agctcccgct gccggaggag ttcctgggct ccgggatgga cttccccgtg
540
ggcaacgtct ccttcttcct cttcttctcg ggccacgtcg cgggcgcggt gatcgcggcg
600
gccgacatgc ggcgcgaggg gcggctggcg ctggcgcgcc tcttcgactc gctcaacgtg
660
ctccaggtgg tcaggctgct cgcgtgcagg ggacactaca ccattgacct ggctgttggc
720
gttggsgcgg gcatcctctt cgacacgctc tccggatggt acttcgacgc caagaacggc
780
gatagcagca acgcaccgga gaagcagtgc cggagctgcc agtgccacaa ggccctcctt
840
tcacactag
849
<210> 265
<211> 282
<212> БЕЛОК
<213> Zea mays
<400> 265
Met Pro Pro Pro Ser Leu Thr Ala Ala Gly Thr Thr Thr Thr Thr Thr
1 5 10 15
Arg Arg Arg Asn Asp Arg Ala Ala Lys Val His Gln Val Leu Gly Glu
20 25 30
Gly Ala Gly Thr Glu Glu Met Gly Ala Val Ala Asp Gly Trp Thr Arg
35 40 45
Pro Glu Trp Cys Ser Ala Ala Gly Val Ala Gly Val Leu Arg Arg His
50 55 60
Pro Ala Pro Ala Leu Phe Gly Cys Gly Leu Leu Leu Phe Met Ala Val
65 70 75 80
Glu Tyr Thr Ile Pro Met Val Lys Pro Asp Ala Pro Pro Leu Asp Leu
85 90 95
Gly Phe Leu Ala Thr Ala Gly Met His Ala Ala Ile Ala Ala Arg Pro
100 105 110
Trp Leu Asn Ser Leu Leu Ala Ala Leu Asn Thr Val Phe Val Ala Met
115 120 125
Gln Ala Ala Tyr Ile Leu Trp Ala Ile Leu Ala Glu Gln Arg Pro Arg
130 135 140
Ala Ala Val Ala Ala Leu Met Met Phe Thr Cys Arg Gly Val Leu Gly
145 150 155 160
Cys Ala Thr Gln Leu Pro Leu Pro Glu Glu Phe Leu Gly Ser Gly Met
165 170 175
Asp Phe Pro Val Gly Asn Val Ser Phe Phe Leu Phe Phe Ser Gly His
180 185 190
Val Ala Gly Ala Val Ile Ala Ala Ala Asp Met Arg Arg Glu Gly Arg
195 200 205
Leu Ala Leu Ala Arg Leu Phe Asp Ser Leu Asn Val Leu Gln Val Val
210 215 220
Arg Leu Leu Ala Cys Arg Gly His Tyr Thr Ile Asp Leu Ala Val Gly
225 230 235 240
Val Gly Ala Gly Ile Leu Phe Asp Thr Leu Ser Gly Trp Tyr Phe Asp
245 250 255
Ala Lys Asn Gly Asp Ser Ser Asn Ala Pro Glu Lys Gln Cys Arg Ser
260 265 270
Cys Gln Cys His Lys Ala Leu Leu Ser His
275 280
<210> 266
<211> 2223
<212> ДНК
<213> Sorghum bicolor
<400> 266
atggggggcg ccgtgttggt cgccatcgcg gcctctatcg gcaacttgct gcagggctgg
60
gacaatgcga caattgctgg agcggtcttg tacataaaga aggaattcaa cttgcagagc
120
gagcccctaa ttgagggcct catcgtggcc atgtccctca ttggggcaac agtcatcacg
180
acgttctctg gggcagtggc cgactctgtt ggtaggaggc ccatgctgat cgcctcggct
240
atcctctact ttgttagtgg gctggtgatg ctctgggcgc caaatgtgta tgtcttgctc
300
cttgcaaggc ttatcgatgg gtttggtatt gggttggccg tcacacttgt tcctttgtac
360
atttctgaga ctgcgccgac ggatattcga gggctgttga acacactacc gcagttcagt
420
ggttcaggag ggatgttcct ttcctactgt atggtgtttg gaatgtccct catgcccaca
480
cctgattgga ggctcatgct tggagttctg tcgatcccat cacttattta ctttggattg
540
actatcttct acttgcctga atcaccaagg tggctcgtga gtaaaggaag gatggctgag
600
gcaaagcgag tgttgcaaag gctgcgggga agggaagatg tctcagggga aatggctctt
660
ctagttgaag gtttgggggt tgggaaagat acacgtattg aagaatacat aattggccct
720
gatgacgagc ttgctgatga agggctggct cccgatccag agaagatcaa attatatgga
780
cctgaagaag gtctatcttg ggttgcccga cctgttcggg gacaaagtgc tcttggaagc
840
gcattaggtc tcatctctcg tcatgggagt atggctagtc agggtaagcc cctcgtggat
900
cctgtggtca ctcttttcgg aagtgttcat gaaaagatgc ctgagataat ggggagcatg
960
aggagcacat tgtttcccaa ctttggcagc atgtttagtg ttgccgaaca gcagcaggtg
1020
aaggctgact gggatgccga gagtcaaagg gagggtgacg attatgcttc agatcatggt
1080
ggcgatgaca ttgaggataa cctccaaagc ccacttattt ctcgtcaagc aacaagtgtg
1140
gaaggaaagg agattgctgc acctcatggt agcataatgg gtgctgtggg aagaagcagt
1200
agcctgcagg gaggggaggc agtaagcagc atgggcattg gtggaggatg gcagttggcg
1260
tggaaatgga ctgagagaga gggcgaagat ggggaaaagg aaggtggctt ccagcgtatt
1320
tatttgcatg aggagggcgt acaaggcagg ggttctatat tgtcattacc aggaggggat
1380
gttcctcctg gtggtgagtt cgtccaggct gcagctcttg tgagtcaacc agctctttac
1440
tcaaaggaac tgctggagca acgtgctgct ggtcctgcga tgatgcatcc atctgaggca
1500
gttgctaaag gtccaagatg ggctgacctg tttgagcctg gagtgaagca tgcactgttt
1560
gttggcatag gaatacagat cctgcaacag tttgctggca tcaatggtgt tctctactac
1620
actcctcaaa ttcttgagca agcaggtgtt ggtgttcttc tgtcgaacat tggccttagc
1680
gcatcttctg catcaattct tattagtgcc ttgacaacct tattgatgct tccaagcatt
1740
ggtattgcaa tgaggctcat ggatatgtct ggaaggaggt ttcttctcct tgcgacaatc
1800
cctatcttga tagttgccct agctatcttg gtcgtggtca atattgtgga tgtgggaacc
1860
atggtgcatg ctgcactctc cacgattagt gtcatagtct atttctgctt ctttgtcatg
1920
gggtttgggc ctattcccaa cattctctgt gcagagatct ttcccaccac cgtccgcggc
1980
atctgcatag ccatctgcgc cttaaccttc tggattggtg acattatcgt gacatacaca
2040
cttcctgtga tgctgaatgc catcgggctc gctggtgtct ttgggatata cgccgtcgtt
2100
tgcatcctgg ctcttgtatt tgtattcatc aaggtgccag agacaaaggg catgcctctc
2160
gaggtcatca ctgagttctt ctccgttgga gcaaagcaag ccaaggaagc cagggaagat
2220
taa
2223
<210> 267
<211> 740
<212> БЕЛОК
<213> Sorghum bicolor
<400> 267
Met Gly Gly Ala Val Leu Val Ala Ile Ala Ala Ser Ile Gly Asn Leu
1 5 10 15
Leu Gln Gly Trp Asp Asn Ala Thr Ile Ala Gly Ala Val Leu Tyr Ile
20 25 30
Lys Lys Glu Phe Asn Leu Gln Ser Glu Pro Leu Ile Glu Gly Leu Ile
35 40 45
Val Ala Met Ser Leu Ile Gly Ala Thr Val Ile Thr Thr Phe Ser Gly
50 55 60
Ala Val Ala Asp Ser Val Gly Arg Arg Pro Met Leu Ile Ala Ser Ala
65 70 75 80
Ile Leu Tyr Phe Val Ser Gly Leu Val Met Leu Trp Ala Pro Asn Val
85 90 95
Tyr Val Leu Leu Leu Ala Arg Leu Ile Asp Gly Phe Gly Ile Gly Leu
100 105 110
Ala Val Thr Leu Val Pro Leu Tyr Ile Ser Glu Thr Ala Pro Thr Asp
115 120 125
Ile Arg Gly Leu Leu Asn Thr Leu Pro Gln Phe Ser Gly Ser Gly Gly
130 135 140
Met Phe Leu Ser Tyr Cys Met Val Phe Gly Met Ser Leu Met Pro Thr
145 150 155 160
Pro Asp Trp Arg Leu Met Leu Gly Val Leu Ser Ile Pro Ser Leu Ile
165 170 175
Tyr Phe Gly Leu Thr Ile Phe Tyr Leu Pro Glu Ser Pro Arg Trp Leu
180 185 190
Val Ser Lys Gly Arg Met Ala Glu Ala Lys Arg Val Leu Gln Arg Leu
195 200 205
Arg Gly Arg Glu Asp Val Ser Gly Glu Met Ala Leu Leu Val Glu Gly
210 215 220
Leu Gly Val Gly Lys Asp Thr Arg Ile Glu Glu Tyr Ile Ile Gly Pro
225 230 235 240
Asp Asp Glu Leu Ala Asp Glu Gly Leu Ala Pro Asp Pro Glu Lys Ile
245 250 255
Lys Leu Tyr Gly Pro Glu Glu Gly Leu Ser Trp Val Ala Arg Pro Val
260 265 270
Arg Gly Gln Ser Ala Leu Gly Ser Ala Leu Gly Leu Ile Ser Arg His
275 280 285
Gly Ser Met Ala Ser Gln Gly Lys Pro Leu Val Asp Pro Val Val Thr
290 295 300
Leu Phe Gly Ser Val His Glu Lys Met Pro Glu Ile Met Gly Ser Met
305 310 315 320
Arg Ser Thr Leu Phe Pro Asn Phe Gly Ser Met Phe Ser Val Ala Glu
325 330 335
Gln Gln Gln Val Lys Ala Asp Trp Asp Ala Glu Ser Gln Arg Glu Gly
340 345 350
Asp Asp Tyr Ala Ser Asp His Gly Gly Asp Asp Ile Glu Asp Asn Leu
355 360 365
Gln Ser Pro Leu Ile Ser Arg Gln Ala Thr Ser Val Glu Gly Lys Glu
370 375 380
Ile Ala Ala Pro His Gly Ser Ile Met Gly Ala Val Gly Arg Ser Ser
385 390 395 400
Ser Leu Gln Gly Gly Glu Ala Val Ser Ser Met Gly Ile Gly Gly Gly
405 410 415
Trp Gln Leu Ala Trp Lys Trp Thr Glu Arg Glu Gly Glu Asp Gly Glu
420 425 430
Lys Glu Gly Gly Phe Gln Arg Ile Tyr Leu His Glu Glu Gly Val Gln
435 440 445
Gly Arg Gly Ser Ile Leu Ser Leu Pro Gly Gly Asp Val Pro Pro Gly
450 455 460
Gly Glu Phe Val Gln Ala Ala Ala Leu Val Ser Gln Pro Ala Leu Tyr
465 470 475 480
Ser Lys Glu Leu Leu Glu Gln Arg Ala Ala Gly Pro Ala Met Met His
485 490 495
Pro Ser Glu Ala Val Ala Lys Gly Pro Arg Trp Ala Asp Leu Phe Glu
500 505 510
Pro Gly Val Lys His Ala Leu Phe Val Gly Ile Gly Ile Gln Ile Leu
515 520 525
Gln Gln Phe Ala Gly Ile Asn Gly Val Leu Tyr Tyr Thr Pro Gln Ile
530 535 540
Leu Glu Gln Ala Gly Val Gly Val Leu Leu Ser Asn Ile Gly Leu Ser
545 550 555 560
Ala Ser Ser Ala Ser Ile Leu Ile Ser Ala Leu Thr Thr Leu Leu Met
565 570 575
Leu Pro Ser Ile Gly Ile Ala Met Arg Leu Met Asp Met Ser Gly Arg
580 585 590
Arg Phe Leu Leu Leu Ala Thr Ile Pro Ile Leu Ile Val Ala Leu Ala
595 600 605
Ile Leu Val Val Val Asn Ile Val Asp Val Gly Thr Met Val His Ala
610 615 620
Ala Leu Ser Thr Ile Ser Val Ile Val Tyr Phe Cys Phe Phe Val Met
625 630 635 640
Gly Phe Gly Pro Ile Pro Asn Ile Leu Cys Ala Glu Ile Phe Pro Thr
645 650 655
Thr Val Arg Gly Ile Cys Ile Ala Ile Cys Ala Leu Thr Phe Trp Ile
660 665 670
Gly Asp Ile Ile Val Thr Tyr Thr Leu Pro Val Met Leu Asn Ala Ile
675 680 685
Gly Leu Ala Gly Val Phe Gly Ile Tyr Ala Val Val Cys Ile Leu Ala
690 695 700
Leu Val Phe Val Phe Ile Lys Val Pro Glu Thr Lys Gly Met Pro Leu
705 710 715 720
Glu Val Ile Thr Glu Phe Phe Ser Val Gly Ala Lys Gln Ala Lys Glu
725 730 735
Ala Arg Glu Asp
740
<210> 268
<211> 2244
<212> ДНК
<213> Zea mays
<400> 268
atggggggcg ccgtgatggt cgccatcgcg gcctctatcg gcaacttgct gcagggctgg
60
gacaatgcga caattgctgg agccgtcctg tacataaaga aggaattcaa cctgcagagc
120
gagcctctga tcgagggcct catcgtcgcc atgtccctca ttggggcaac agtcatcacg
180
acgttctccg gggcagcggc cgactgcgtt ggtaggaggc ccatgctggt cgcctcggct
240
gtcctctact tcgtcagtgg gctggtgatg ctttgggcgc caagtgtgta catcttgctc
300
ctcgcaaggc tcattgatgg gttcggtatc ggtttggcgg tcacacttgt tcctctctac
360
atctccgaga ctgcaccgac agacattcgt gggctgttga acacgttgcc gcagttcagt
420
gggtcaggag ggatgttcct ctcctactgc atggtgtttg ggatgtccct catgcccaaa
480
cctgattgga ggctcatgct tggagttctg tcgatcccgt cacttattta ctttggactg
540
actgtcttct acttgcctga atcaccaagg tggcttgtga gcaaaggaag gatggcggag
600
gcgaagagag tgttgcaaag gctgcgggga agagaagatg tctcagggga gatggctctt
660
ctagttgaag gtttgggggt cggtaaagat acacgtattg aagaatacat aattggtccc
720
gatgatgaac ttgctgatga agggctggct ccagatccag agaagatcaa actatatgga
780
cctgaagaag gcctatcttg ggttgcccga cctgttcggg gacaaagtgc tcttggaagc
840
gcgttaggtc tcatctctcg tcatgggagt atggcggcta gtcagggtaa gcccctcgtg
900
gatcctatgg tcactctttt cggaagtgtt catgaaaaga tgcctgagat catggggagc
960
atgaggagca cattgtttcc caactttggc agcatgttta gtgttgccga ccagcagcag
1020
gtgaaagctg actgggacgc cgagagtcaa agggaaggtg aagattatgc ttcggatcat
1080
ggtggcgatg acatcgagga taacctccaa agcccactta tttctcgtca ggcaacaagt
1140
gtggaaggaa aggagatcgc tgcacctcat ggtagcatat tgggtgctgt gggaaggagc
1200
agtagcttgc agggagggga ggcagtaagc agcatgggca ttggcggagg atggcagttg
1260
gcgtggaaat ggaccgagag agagggcgaa gatgggcaaa aggaaggtgg cttccagcgt
1320
atttacttgc atgaggaggg cgtacaaggc aacaggggtt ctatattgtc attaccaggc
1380
ggggatgttc ctcctggtgg tgagttcatc caggctgcag ctcttgtgag ccaaccagct
1440
ctttactcta aggaactgct ggagcaacgt gctgctggtc ctgcgatgat gcatccatct
1500
gaagcagtta ctaaaggtcc aagatgggcc gacctatttg agcctggggt gaagcatgca
1560
ctgtttgttg gcataggaat acagatcctg caacagtttg ctggcatcaa cggcgttctc
1620
tactacactc ctcaaattct tgagcaagca ggcgttggtg ttcttctgtc gaacctcggc
1680
cttaacgctt cttcggcatc aatcctcatt agcgccctga cgaccttact gatgctccca
1740
agcatcggca ttgcgatgag gctcatggat atgtccggaa ggaggtttct cctcctcgcg
1800
acgatcccag tcctaatagt cgcgctactc gtcctggtgg tgtccaacat cgtggacgtg
1860
ggggacgtgg cgcacgcggc gctctccacg gccagcgtca tagtctactt ctgcttcttc
1920
gtcatggggt tcgggcccgt ccccaacatc ctctgcgcag agatcttccc caccacggtc
1980
cgcggtgtct gcatcgccat ctgcgccctg gccttctggc tcggtgacat catcgtgacg
2040
tacactctcc ccgtgatgct gaacgtcgtc gggctcgccg gcgtctttgg ggtgtacgcc
2100
gtcgtgtgcg tcctagccct cgcgttcgtg ttcgtcaagg tgcccgagac gaagggcatg
2160
cctctcgagg tcatcaccga gttcttctcc gttggggcaa agcaagccaa ggaagaggag
2220
gaggaggagg ccagggaagg ttga
2244
<210> 269
<211> 747
<212> БЕЛОК
<213> Zea mays
<400> 269
Met Gly Gly Ala Val Met Val Ala Ile Ala Ala Ser Ile Gly Asn Leu
1 5 10 15
Leu Gln Gly Trp Asp Asn Ala Thr Ile Ala Gly Ala Val Leu Tyr Ile
20 25 30
Lys Lys Glu Phe Asn Leu Gln Ser Glu Pro Leu Ile Glu Gly Leu Ile
35 40 45
Val Ala Met Ser Leu Ile Gly Ala Thr Val Ile Thr Thr Phe Ser Gly
50 55 60
Ala Ala Ala Asp Cys Val Gly Arg Arg Pro Met Leu Val Ala Ser Ala
65 70 75 80
Val Leu Tyr Phe Val Ser Gly Leu Val Met Leu Trp Ala Pro Ser Val
85 90 95
Tyr Ile Leu Leu Leu Ala Arg Leu Ile Asp Gly Phe Gly Ile Gly Leu
100 105 110
Ala Val Thr Leu Val Pro Leu Tyr Ile Ser Glu Thr Ala Pro Thr Asp
115 120 125
Ile Arg Gly Leu Leu Asn Thr Leu Pro Gln Phe Ser Gly Ser Gly Gly
130 135 140
Met Phe Leu Ser Tyr Cys Met Val Phe Gly Met Ser Leu Met Pro Lys
145 150 155 160
Pro Asp Trp Arg Leu Met Leu Gly Val Leu Ser Ile Pro Ser Leu Ile
165 170 175
Tyr Phe Gly Leu Thr Val Phe Tyr Leu Pro Glu Ser Pro Arg Trp Leu
180 185 190
Val Ser Lys Gly Arg Met Ala Glu Ala Lys Arg Val Leu Gln Arg Leu
195 200 205
Arg Gly Arg Glu Asp Val Ser Gly Glu Met Ala Leu Leu Val Glu Gly
210 215 220
Leu Gly Val Gly Lys Asp Thr Arg Ile Glu Glu Tyr Ile Ile Gly Pro
225 230 235 240
Asp Asp Glu Leu Ala Asp Glu Gly Leu Ala Pro Asp Pro Glu Lys Ile
245 250 255
Lys Leu Tyr Gly Pro Glu Glu Gly Leu Ser Trp Val Ala Arg Pro Val
260 265 270
Arg Gly Gln Ser Ala Leu Gly Ser Ala Leu Gly Leu Ile Ser Arg His
275 280 285
Gly Ser Met Ala Ala Ser Gln Gly Lys Pro Leu Val Asp Pro Met Val
290 295 300
Thr Leu Phe Gly Ser Val His Glu Lys Met Pro Glu Ile Met Gly Ser
305 310 315 320
Met Arg Ser Thr Leu Phe Pro Asn Phe Gly Ser Met Phe Ser Val Ala
325 330 335
Asp Gln Gln Gln Val Lys Ala Asp Trp Asp Ala Glu Ser Gln Arg Glu
340 345 350
Gly Glu Asp Tyr Ala Ser Asp His Gly Gly Asp Asp Ile Glu Asp Asn
355 360 365
Leu Gln Ser Pro Leu Ile Ser Arg Gln Ala Thr Ser Val Glu Gly Lys
370 375 380
Glu Ile Ala Ala Pro His Gly Ser Ile Leu Gly Ala Val Gly Arg Ser
385 390 395 400
Ser Ser Leu Gln Gly Gly Glu Ala Val Ser Ser Met Gly Ile Gly Gly
405 410 415
Gly Trp Gln Leu Ala Trp Lys Trp Thr Glu Arg Glu Gly Glu Asp Gly
420 425 430
Gln Lys Glu Gly Gly Phe Gln Arg Ile Tyr Leu His Glu Glu Gly Val
435 440 445
Gln Gly Asn Arg Gly Ser Ile Leu Ser Leu Pro Gly Gly Asp Val Pro
450 455 460
Pro Gly Gly Glu Phe Ile Gln Ala Ala Ala Leu Val Ser Gln Pro Ala
465 470 475 480
Leu Tyr Ser Lys Glu Leu Leu Glu Gln Arg Ala Ala Gly Pro Ala Met
485 490 495
Met His Pro Ser Glu Ala Val Thr Lys Gly Pro Arg Trp Ala Asp Leu
500 505 510
Phe Glu Pro Gly Val Lys His Ala Leu Phe Val Gly Ile Gly Ile Gln
515 520 525
Ile Leu Gln Gln Phe Ala Gly Ile Asn Gly Val Leu Tyr Tyr Thr Pro
530 535 540
Gln Ile Leu Glu Gln Ala Gly Val Gly Val Leu Leu Ser Asn Leu Gly
545 550 555 560
Leu Asn Ala Ser Ser Ala Ser Ile Leu Ile Ser Ala Leu Thr Thr Leu
565 570 575
Leu Met Leu Pro Ser Ile Gly Ile Ala Met Arg Leu Met Asp Met Ser
580 585 590
Gly Arg Arg Phe Leu Leu Leu Ala Thr Ile Pro Val Leu Ile Val Ala
595 600 605
Leu Leu Val Leu Val Val Ser Asn Ile Val Asp Val Gly Asp Val Ala
610 615 620
His Ala Ala Leu Ser Thr Ala Ser Val Ile Val Tyr Phe Cys Phe Phe
625 630 635 640
Val Met Gly Phe Gly Pro Val Pro Asn Ile Leu Cys Ala Glu Ile Phe
645 650 655
Pro Thr Thr Val Arg Gly Val Cys Ile Ala Ile Cys Ala Leu Ala Phe
660 665 670
Trp Leu Gly Asp Ile Ile Val Thr Tyr Thr Leu Pro Val Met Leu Asn
675 680 685
Val Val Gly Leu Ala Gly Val Phe Gly Val Tyr Ala Val Val Cys Val
690 695 700
Leu Ala Leu Ala Phe Val Phe Val Lys Val Pro Glu Thr Lys Gly Met
705 710 715 720
Pro Leu Glu Val Ile Thr Glu Phe Phe Ser Val Gly Ala Lys Gln Ala
725 730 735
Lys Glu Glu Glu Glu Glu Glu Ala Arg Glu Gly
740 745
<210> 270
<211> 2238
<212> ДНК
<213> Sorghum bicolor
<400> 270
atgtcggggg ctgttcttgt cgccatcgcc gcctccatcg gcaatctgtt gcaggggtgg
60
gacaatgcca ccatcgcagg tgctgttctg tacataaaga aggaattcca attacaaaat
120
gagcccactg tggaggggct gattgtggcc atgtcactta ttggtgccac catcatcact
180
acattctctg ggccagtatc agactggatc ggccgccgcc ctatgctcat cctctcgtca
240
attctgtact ttctcagcag cctcatcatg ctatggtccc ctaatgtcta tgtcctgctg
300
ctggcacgcc tcgtagacgg attcggtatt ggcttggctg tcacgcttgt gcctttgtac
360
atttcagaaa cagcccctcc agagattaga ggtttgctga atacactgcc acagttcagt
420
ggatcaggag ggatgttctt gtcatactgc atggtgtttg ggatgtcact gttgccatca
480
cctgattgga gaattatgct tggggtgctc gcgatacctt cattgttctt ctttggattg
540
acaatatttt accttcctga atccccaaga tggcttgtta gcaaaggtcg gatggcagag
600
gcaaagaagg tgttgcaaaa attacgcagc aaagaagatg tctcaggtga attgtccctt
660
cttgttgaag ggttggaggt tggaggagac acttcgattg aagagtacat cattggccct
720
gccactgacg cagccgatga tcatgttact gatggtgata aggaacaaat cacactttat
780
gggcctgaag aaggccagtc atggattgct cgaccttcca agggacccag catgcttgga
840
agtgtacttt ctctcgcatc tcgtcatggc agcttggtga accagagtgt accccttatg
900
gatccgattg tgacactttt tgggagtgtc catgagaata tgcctcaagc tggaggaagt
960
atgaggagca cattgtttcc aaactttgga agtatgttca gtgtcacaga tcagcatgcc
1020
aaaaatgagc agtgggacga agagaatctt cacagggacg atgaggagta tgcatctgat
1080
ggtgcaggag gtgattatga ggacaatctc cacagtccat tgctgtccag gcagacaaca
1140
agtgcggaag ggaaggacat tgtgcaccat ggtcaccgtg gaagttcttt gagcatgaga
1200
aggcaaagcc tcttggggga ggctggagag ggtgtgagca gcactgatat tggtggggga
1260
tggcagcttg catggaaatg gtcagagaag gaaggtgagg atggtaagaa ggaaggtggt
1320
ttcaaaagag tctacttgca ccaagaggga gttcctggct caagaatggg ctcaattgtt
1380
tcacttcctg gtggtggcga tgttcatgag ggtggcgagt ttgtacatgc tgctgcttta
1440
gtaagccagt cagcactttt ctcgaaggat cttaccgaac cacgcatgtc tggtgctgcc
1500
atgattaacg catccgaggt agccgccaaa ggttcaagct ggaaagattt gtttgaacct
1560
ggtgtgaggc gtgccctgtt agtcggtgtt ggaattcaga tccttcaaca gtttgctgga
1620
atcaatggtg ttctgtacta taccccacaa attctcgagc aagctggcgt ggcagttctt
1680
ctttccaatc ttggtctcag ctcagcatca gcatctatct tgatcagttc tctcactacc
1740
ttactgatgc ttcctagcat tggcttagcc atgagactta tggatctttc tggaagaagg
1800
tttttgctgc taggcacaat tccaatcttg atagcatctt tagttatcct ggtcgtgtcc
1860
aatgtgattg acctgggtac agtggcccat gctgcgctct ccacagtcag tgtcatcatc
1920
tacttctgct gctttgtcat gggatttggt cccatcccca acattctatg tgcagagatc
1980
tttccaacca gggttcgcgg tctctgcatt gccatctgtg ccttgacgtt ttggattgga
2040
gacatcattg tcacctacag ccttcctgta atgctgaatg ctattggact agcaggtgtt
2100
tttggcatat atgcagtcgt atgcttgatt gcctttgtgt ttgtcttcct taaggttcct
2160
gagacaaagg gaatgcccct tgaagtcatc actgagttct ttgcagttgg tgcgaagcaa
2220
gcggctgcaa aagcctaa
2238
<210> 271
<211> 745
<212> БЕЛОК
<213> Sorghum bicolor
<400> 271
Met Ser Gly Ala Val Leu Val Ala Ile Ala Ala Ser Ile Gly Asn Leu
1 5 10 15
Leu Gln Gly Trp Asp Asn Ala Thr Ile Ala Gly Ala Val Leu Tyr Ile
20 25 30
Lys Lys Glu Phe Gln Leu Gln Asn Glu Pro Thr Val Glu Gly Leu Ile
35 40 45
Val Ala Met Ser Leu Ile Gly Ala Thr Ile Ile Thr Thr Phe Ser Gly
50 55 60
Pro Val Ser Asp Trp Ile Gly Arg Arg Pro Met Leu Ile Leu Ser Ser
65 70 75 80
Ile Leu Tyr Phe Leu Ser Ser Leu Ile Met Leu Trp Ser Pro Asn Val
85 90 95
Tyr Val Leu Leu Leu Ala Arg Leu Val Asp Gly Phe Gly Ile Gly Leu
100 105 110
Ala Val Thr Leu Val Pro Leu Tyr Ile Ser Glu Thr Ala Pro Pro Glu
115 120 125
Ile Arg Gly Leu Leu Asn Thr Leu Pro Gln Phe Ser Gly Ser Gly Gly
130 135 140
Met Phe Leu Ser Tyr Cys Met Val Phe Gly Met Ser Leu Leu Pro Ser
145 150 155 160
Pro Asp Trp Arg Ile Met Leu Gly Val Leu Ala Ile Pro Ser Leu Phe
165 170 175
Phe Phe Gly Leu Thr Ile Phe Tyr Leu Pro Glu Ser Pro Arg Trp Leu
180 185 190
Val Ser Lys Gly Arg Met Ala Glu Ala Lys Lys Val Leu Gln Lys Leu
195 200 205
Arg Ser Lys Glu Asp Val Ser Gly Glu Leu Ser Leu Leu Val Glu Gly
210 215 220
Leu Glu Val Gly Gly Asp Thr Ser Ile Glu Glu Tyr Ile Ile Gly Pro
225 230 235 240
Ala Thr Asp Ala Ala Asp Asp His Val Thr Asp Gly Asp Lys Glu Gln
245 250 255
Ile Thr Leu Tyr Gly Pro Glu Glu Gly Gln Ser Trp Ile Ala Arg Pro
260 265 270
Ser Lys Gly Pro Ser Met Leu Gly Ser Val Leu Ser Leu Ala Ser Arg
275 280 285
His Gly Ser Leu Val Asn Gln Ser Val Pro Leu Met Asp Pro Ile Val
290 295 300
Thr Leu Phe Gly Ser Val His Glu Asn Met Pro Gln Ala Gly Gly Ser
305 310 315 320
Met Arg Ser Thr Leu Phe Pro Asn Phe Gly Ser Met Phe Ser Val Thr
325 330 335
Asp Gln His Ala Lys Asn Glu Gln Trp Asp Glu Glu Asn Leu His Arg
340 345 350
Asp Asp Glu Glu Tyr Ala Ser Asp Gly Ala Gly Gly Asp Tyr Glu Asp
355 360 365
Asn Leu His Ser Pro Leu Leu Ser Arg Gln Thr Thr Ser Ala Glu Gly
370 375 380
Lys Asp Ile Val His His Gly His Arg Gly Ser Ser Leu Ser Met Arg
385 390 395 400
Arg Gln Ser Leu Leu Gly Glu Ala Gly Glu Gly Val Ser Ser Thr Asp
405 410 415
Ile Gly Gly Gly Trp Gln Leu Ala Trp Lys Trp Ser Glu Lys Glu Gly
420 425 430
Glu Asp Gly Lys Lys Glu Gly Gly Phe Lys Arg Val Tyr Leu His Gln
435 440 445
Glu Gly Val Pro Gly Ser Arg Met Gly Ser Ile Val Ser Leu Pro Gly
450 455 460
Gly Gly Asp Val His Glu Gly Gly Glu Phe Val His Ala Ala Ala Leu
465 470 475 480
Val Ser Gln Ser Ala Leu Phe Ser Lys Asp Leu Thr Glu Pro Arg Met
485 490 495
Ser Gly Ala Ala Met Ile Asn Ala Ser Glu Val Ala Ala Lys Gly Ser
500 505 510
Ser Trp Lys Asp Leu Phe Glu Pro Gly Val Arg Arg Ala Leu Leu Val
515 520 525
Gly Val Gly Ile Gln Ile Leu Gln Gln Phe Ala Gly Ile Asn Gly Val
530 535 540
Leu Tyr Tyr Thr Pro Gln Ile Leu Glu Gln Ala Gly Val Ala Val Leu
545 550 555 560
Leu Ser Asn Leu Gly Leu Ser Ser Ala Ser Ala Ser Ile Leu Ile Ser
565 570 575
Ser Leu Thr Thr Leu Leu Met Leu Pro Ser Ile Gly Leu Ala Met Arg
580 585 590
Leu Met Asp Leu Ser Gly Arg Arg Phe Leu Leu Leu Gly Thr Ile Pro
595 600 605
Ile Leu Ile Ala Ser Leu Val Ile Leu Val Val Ser Asn Val Ile Asp
610 615 620
Leu Gly Thr Val Ala His Ala Ala Leu Ser Thr Val Ser Val Ile Ile
625 630 635 640
Tyr Phe Cys Cys Phe Val Met Gly Phe Gly Pro Ile Pro Asn Ile Leu
645 650 655
Cys Ala Glu Ile Phe Pro Thr Arg Val Arg Gly Leu Cys Ile Ala Ile
660 665 670
Cys Ala Leu Thr Phe Trp Ile Gly Asp Ile Ile Val Thr Tyr Ser Leu
675 680 685
Pro Val Met Leu Asn Ala Ile Gly Leu Ala Gly Val Phe Gly Ile Tyr
690 695 700
Ala Val Val Cys Leu Ile Ala Phe Val Phe Val Phe Leu Lys Val Pro
705 710 715 720
Glu Thr Lys Gly Met Pro Leu Glu Val Ile Thr Glu Phe Phe Ala Val
725 730 735
Gly Ala Lys Gln Ala Ala Ala Lys Ala
740 745
<210> 272
<211> 2238
<212> ДНК
<213> Zea mays
<400> 272
atgtcggggg ctgttcttgt cgccatagtc gcctccatcg gcaatctatt gcaggggtgg
60
gacaatgcca ccatcgcagc tgctgttctg tatataaaga aggaatttca attgcaaaat
120
gagcccactg tggagggact aattgtgtca atgtcactta tcggcgccac catcgttact
180
acattctccg ggccattatc agactcgatt ggccgacgcc ctatgcttat tctctcttca
240
attctgtact tcttcagcgg cctcatcatg ctatggtctc ctaatgtcta tgtcctgctg
300
ttggcacgct tcgtagatgg atttggtatt ggcttggctg tcacgcttgt gcctttgtac
360
atttcagaaa tagccccttc ggagattaga ggtttgctga atacactacc acaattcagt
420
ggatcaggag gaatgttctt gtcatactgc atggtgtttg ggatgtccct gtcgccatca
480
cccgattgga gaattatgct tggtgtgctc gcgatacctt cattgttctt ctttggtttg
540
acaatatttt atcttcctga atctccaaga tggctcgtta gcaaaggtcg gatggcagag
600
gcaaaaaagg tgttgcaaaa gttacggggg aaagacgatg tctcaggtga attgtccctt
660
cttctcgaag ggttggaggt tggaggagac acttccattg aagagtacat cattggacct
720
gccaccgagg cagccgatga tcttgttact gacggtgata aggaacaaat cacactttat
780
gggcctgaag aaggccagtc atggattgct cgaccttcca agggacccag catgcttgga
840
agtgtgcttt ctcttgcatc tcgtcatggg agcatggtga accagagtgt accccttatg
900
gatccgattg tgacactttt tggtagtgtc catgagaata tgcctcaagc tggaggaagt
960
atgaggagca cattgtttcc aaactttgga agtatgttca gtgtcacaga tcagcatgcc
1020
aaaaatgagc agtgggatga agagaatctt catagggatg acgaggagta cgcatctgat
1080
ggtgcaggag gtgactatga ggacaatctc catagcccat tgctgtccag gcaggcaaca
1140
ggtgcggaag ggaaggacat tgtgcaccat ggtcaccgtg gaagtgcttt gagcatgaga
1200
aggcaaagcc tcttagggga gggtggagat ggtgtgagca gcactgatat cggtggggga
1260
tggcagcttg cttggaaatg gtcagagaag gaaggtgaga atggtagaaa ggaaggtggt
1320
ttcaaaagag tctacttgca ccaagaggga gttcctggct caagaagggg ctcaattgtt
1380
tcacttcccg gtggtggcga tgttcttgag ggtagtgagt ttgtacatgc tgctgcttta
1440
gtaagtcagt cagcactttt ctcaaagggt cttgctgaac cacgcatgtc agatgctgcc
1500
atggttcacc catctgaggt agctgccaaa ggttcacgtt ggaaagattt gtttgaacct
1560
ggagtgaggc gtgccctgtt agtcggtgtt ggaattcaga tccttcaaca gtttgctgga
1620
ataaacggtg ttctgtacta taccccacaa attcttgagc aagctggtgt ggcagttatt
1680
ctttccaaat ttggtctcag ctcggcatca gcatccatct tgatcagttc tctcactacc
1740
ttactaatgc ttccttgcat tggctttgcc atgctgctta tggatctttc cggaagaagg
1800
tttttgctgc taggcacaat tccaatcttg atagcatctc tagttatcct ggttgtgtcc
1860
aatctaattg atttgggtac actagcccat gctttgctct ccaccgtcag tgttatcgtc
1920
tacttctgct gcttcgttat gggatttggt cccatcccca acattttatg tgcagagatc
1980
tttccaacca gggttcgtgg cctctgtatt gccatttgtg cctttacatt ctggatcgga
2040
gatatcatcg tcacctacag ccttcctgtg atgctgaatg ctattggact ggcgggtgtt
2100
ttcagcatat atgcagtcgt atgcttgatt tcctttgtgt tcgtcttcct taaggtccct
2160
gagacaaagg ggatgcccct tgaggttatt accgaattct ttgcagttgg tgcgaagcaa
2220
gcggctgcaa aagcctaa
2238
<210> 273
<211> 745
<212> БЕЛОК
<213> Zea mays
<400> 273
Met Ser Gly Ala Val Leu Val Ala Ile Val Ala Ser Ile Gly Asn Leu
1 5 10 15
Leu Gln Gly Trp Asp Asn Ala Thr Ile Ala Ala Ala Val Leu Tyr Ile
20 25 30
Lys Lys Glu Phe Gln Leu Gln Asn Glu Pro Thr Val Glu Gly Leu Ile
35 40 45
Val Ser Met Ser Leu Ile Gly Ala Thr Ile Val Thr Thr Phe Ser Gly
50 55 60
Pro Leu Ser Asp Ser Ile Gly Arg Arg Pro Met Leu Ile Leu Ser Ser
65 70 75 80
Ile Leu Tyr Phe Phe Ser Gly Leu Ile Met Leu Trp Ser Pro Asn Val
85 90 95
Tyr Val Leu Leu Leu Ala Arg Phe Val Asp Gly Phe Gly Ile Gly Leu
100 105 110
Ala Val Thr Leu Val Pro Leu Tyr Ile Ser Glu Ile Ala Pro Ser Glu
115 120 125
Ile Arg Gly Leu Leu Asn Thr Leu Pro Gln Phe Ser Gly Ser Gly Gly
130 135 140
Met Phe Leu Ser Tyr Cys Met Val Phe Gly Met Ser Leu Ser Pro Ser
145 150 155 160
Pro Asp Trp Arg Ile Met Leu Gly Val Leu Ala Ile Pro Ser Leu Phe
165 170 175
Phe Phe Gly Leu Thr Ile Phe Tyr Leu Pro Glu Ser Pro Arg Trp Leu
180 185 190
Val Ser Lys Gly Arg Met Ala Glu Ala Lys Lys Val Leu Gln Lys Leu
195 200 205
Arg Gly Lys Asp Asp Val Ser Gly Glu Leu Ser Leu Leu Leu Glu Gly
210 215 220
Leu Glu Val Gly Gly Asp Thr Ser Ile Glu Glu Tyr Ile Ile Gly Pro
225 230 235 240
Ala Thr Glu Ala Ala Asp Asp Leu Val Thr Asp Gly Asp Lys Glu Gln
245 250 255
Ile Thr Leu Tyr Gly Pro Glu Glu Gly Gln Ser Trp Ile Ala Arg Pro
260 265 270
Ser Lys Gly Pro Ser Met Leu Gly Ser Val Leu Ser Leu Ala Ser Arg
275 280 285
His Gly Ser Met Val Asn Gln Ser Val Pro Leu Met Asp Pro Ile Val
290 295 300
Thr Leu Phe Gly Ser Val His Glu Asn Met Pro Gln Ala Gly Gly Ser
305 310 315 320
Met Arg Ser Thr Leu Phe Pro Asn Phe Gly Ser Met Phe Ser Val Thr
325 330 335
Asp Gln His Ala Lys Asn Glu Gln Trp Asp Glu Glu Asn Leu His Arg
340 345 350
Asp Asp Glu Glu Tyr Ala Ser Asp Gly Ala Gly Gly Asp Tyr Glu Asp
355 360 365
Asn Leu His Ser Pro Leu Leu Ser Arg Gln Ala Thr Gly Ala Glu Gly
370 375 380
Lys Asp Ile Val His His Gly His Arg Gly Ser Ala Leu Ser Met Arg
385 390 395 400
Arg Gln Ser Leu Leu Gly Glu Gly Gly Asp Gly Val Ser Ser Thr Asp
405 410 415
Ile Gly Gly Gly Trp Gln Leu Ala Trp Lys Trp Ser Glu Lys Glu Gly
420 425 430
Glu Asn Gly Arg Lys Glu Gly Gly Phe Lys Arg Val Tyr Leu His Gln
435 440 445
Glu Gly Val Pro Gly Ser Arg Arg Gly Ser Ile Val Ser Leu Pro Gly
450 455 460
Gly Gly Asp Val Leu Glu Gly Ser Glu Phe Val His Ala Ala Ala Leu
465 470 475 480
Val Ser Gln Ser Ala Leu Phe Ser Lys Gly Leu Ala Glu Pro Arg Met
485 490 495
Ser Asp Ala Ala Met Val His Pro Ser Glu Val Ala Ala Lys Gly Ser
500 505 510
Arg Trp Lys Asp Leu Phe Glu Pro Gly Val Arg Arg Ala Leu Leu Val
515 520 525
Gly Val Gly Ile Gln Ile Leu Gln Gln Phe Ala Gly Ile Asn Gly Val
530 535 540
Leu Tyr Tyr Thr Pro Gln Ile Leu Glu Gln Ala Gly Val Ala Val Ile
545 550 555 560
Leu Ser Lys Phe Gly Leu Ser Ser Ala Ser Ala Ser Ile Leu Ile Ser
565 570 575
Ser Leu Thr Thr Leu Leu Met Leu Pro Cys Ile Gly Phe Ala Met Leu
580 585 590
Leu Met Asp Leu Ser Gly Arg Arg Phe Leu Leu Leu Gly Thr Ile Pro
595 600 605
Ile Leu Ile Ala Ser Leu Val Ile Leu Val Val Ser Asn Leu Ile Asp
610 615 620
Leu Gly Thr Leu Ala His Ala Leu Leu Ser Thr Val Ser Val Ile Val
625 630 635 640
Tyr Phe Cys Cys Phe Val Met Gly Phe Gly Pro Ile Pro Asn Ile Leu
645 650 655
Cys Ala Glu Ile Phe Pro Thr Arg Val Arg Gly Leu Cys Ile Ala Ile
660 665 670
Cys Ala Phe Thr Phe Trp Ile Gly Asp Ile Ile Val Thr Tyr Ser Leu
675 680 685
Pro Val Met Leu Asn Ala Ile Gly Leu Ala Gly Val Phe Ser Ile Tyr
690 695 700
Ala Val Val Cys Leu Ile Ser Phe Val Phe Val Phe Leu Lys Val Pro
705 710 715 720
Glu Thr Lys Gly Met Pro Leu Glu Val Ile Thr Glu Phe Phe Ala Val
725 730 735
Gly Ala Lys Gln Ala Ala Ala Lys Ala
740 745
<210> 274
<211> 1464
<212> ДНК
<213> Sorghum bicolor
<400> 274
atggtgccgg accagtggta cgacaccaac ggcgtctgga caggctccgc caccacgctc
60
cccgacggcc gcctcgccat gctctacacc ggctccacca acgcctccgt gcaggtgcag
120
tgcctcgccg tccccgccga cgacgccgac ccgctgctca ccaactggac caagtacgag
180
ggcaacccgg tcctgtaccc gccgccgggg atcgggccca aggacttccg tgaccccacc
240
acggcgtggt tcgacccgtc ggacaacacc tggcgcatcg tcatcgggtc caaggacgac
300
gccgagggcg accacgccgg catcgccgtc gtctaccgga ccaaggactt cgtcagcttc
360
gagctcctcc ccggcctcct ccaccgcgtc gcgaggacgg ggatgtggga gtgcatcgac
420
ttctaccccg tcgccacccg cggcaaggcg tccgggaacg gcgtcgacat gtccgacgcc
480
ttcggcaaga acggcgccat tgttggggac gtcgtgcacg ttatgaaggc cagcatggac
540
gacgaccgcc atgactacta cgcgctcggg aggtacgatg cggccaccaa cgagtggacg
600
ccgctcgacg ccgagaagga cgtcggcatc gggctccggt atgactgggg caagttttac
660
gcgtccaaga ccttctatga ccccgccaag cgccgccgcg tgctctgggg atgggtcggc
720
gagaccgact cggagcgcgc tgacgtctcc aagggatggg catcgttgca gggtatcccc
780
cggacggtgc tgctggacac caagacgggc agcaacctgc tgcagtggcc cgtggaggaa
840
gcggagacgc tgcgcaccaa ctccacggac ctcagcggca tcaccatcga ctacggctcg
900
gcgttcccgc tcaacctccg gcgcgccacg cagctggaca tcgaggcgga gttccagctc
960
gaccgccgcg ccgtcatgtc gctcaacgag gccgacgtgg ggtacaactg cagcacgagc
1020
ggtggcgccg cggcccgcgg tgccctcggc cccttcggcc tgctcgtcct cgccgaccag
1080
cacctgcgcg agcagacggc cgtctacttc tacgtggcca agggcctgga cggctccctc
1140
accacgcact tttgccagga cgagtcccgg tcctccagcg ccaacgacat cgtcaagcgc
1200
gtcgtcggca gctccgtccc cgtgctggac gacgagacca cgctctcgct ccgcgtgctc
1260
gtcgaccact ccatcgtcga gagcttcgcg cagggcggaa ggtcgacggc aacctcgcgc
1320
gtctacccca ccgaggccat ctacgccaac gccggcgtgt tcctcttcaa caacgccacc
1380
gccgcgcgcg tcaccgccaa gaagctcgtc gtccacgaga tggactcatc ctacaaccac
1440
gactacatgg tcacggacat ctga
1464
<210> 275
<211> 487
<212> БЕЛОК
<213> Sorghum bicolor
<400> 275
Met Val Pro Asp Gln Trp Tyr Asp Thr Asn Gly Val Trp Thr Gly Ser
1 5 10 15
Ala Thr Thr Leu Pro Asp Gly Arg Leu Ala Met Leu Tyr Thr Gly Ser
20 25 30
Thr Asn Ala Ser Val Gln Val Gln Cys Leu Ala Val Pro Ala Asp Asp
35 40 45
Ala Asp Pro Leu Leu Thr Asn Trp Thr Lys Tyr Glu Gly Asn Pro Val
50 55 60
Leu Tyr Pro Pro Pro Gly Ile Gly Pro Lys Asp Phe Arg Asp Pro Thr
65 70 75 80
Thr Ala Trp Phe Asp Pro Ser Asp Asn Thr Trp Arg Ile Val Ile Gly
85 90 95
Ser Lys Asp Asp Ala Glu Gly Asp His Ala Gly Ile Ala Val Val Tyr
100 105 110
Arg Thr Lys Asp Phe Val Ser Phe Glu Leu Leu Pro Gly Leu Leu His
115 120 125
Arg Val Ala Arg Thr Gly Met Trp Glu Cys Ile Asp Phe Tyr Pro Val
130 135 140
Ala Thr Arg Gly Lys Ala Ser Gly Asn Gly Val Asp Met Ser Asp Ala
145 150 155 160
Phe Gly Lys Asn Gly Ala Ile Val Gly Asp Val Val His Val Met Lys
165 170 175
Ala Ser Met Asp Asp Asp Arg His Asp Tyr Tyr Ala Leu Gly Arg Tyr
180 185 190
Asp Ala Ala Thr Asn Glu Trp Thr Pro Leu Asp Ala Glu Lys Asp Val
195 200 205
Gly Ile Gly Leu Arg Tyr Asp Trp Gly Lys Phe Tyr Ala Ser Lys Thr
210 215 220
Phe Tyr Asp Pro Ala Lys Arg Arg Arg Val Leu Trp Gly Trp Val Gly
225 230 235 240
Glu Thr Asp Ser Glu Arg Ala Asp Val Ser Lys Gly Trp Ala Ser Leu
245 250 255
Gln Gly Ile Pro Arg Thr Val Leu Leu Asp Thr Lys Thr Gly Ser Asn
260 265 270
Leu Leu Gln Trp Pro Val Glu Glu Ala Glu Thr Leu Arg Thr Asn Ser
275 280 285
Thr Asp Leu Ser Gly Ile Thr Ile Asp Tyr Gly Ser Ala Phe Pro Leu
290 295 300
Asn Leu Arg Arg Ala Thr Gln Leu Asp Ile Glu Ala Glu Phe Gln Leu
305 310 315 320
Asp Arg Arg Ala Val Met Ser Leu Asn Glu Ala Asp Val Gly Tyr Asn
325 330 335
Cys Ser Thr Ser Gly Gly Ala Ala Ala Arg Gly Ala Leu Gly Pro Phe
340 345 350
Gly Leu Leu Val Leu Ala Asp Gln His Leu Arg Glu Gln Thr Ala Val
355 360 365
Tyr Phe Tyr Val Ala Lys Gly Leu Asp Gly Ser Leu Thr Thr His Phe
370 375 380
Cys Gln Asp Glu Ser Arg Ser Ser Ser Ala Asn Asp Ile Val Lys Arg
385 390 395 400
Val Val Gly Ser Ser Val Pro Val Leu Asp Asp Glu Thr Thr Leu Ser
405 410 415
Leu Arg Val Leu Val Asp His Ser Ile Val Glu Ser Phe Ala Gln Gly
420 425 430
Gly Arg Ser Thr Ala Thr Ser Arg Val Tyr Pro Thr Glu Ala Ile Tyr
435 440 445
Ala Asn Ala Gly Val Phe Leu Phe Asn Asn Ala Thr Ala Ala Arg Val
450 455 460
Thr Ala Lys Lys Leu Val Val His Glu Met Asp Ser Ser Tyr Asn His
465 470 475 480
Asp Tyr Met Val Thr Asp Ile
485
<210> 276
<211> 638
<212> БЕЛОК
<213> Sorghum bicolor
<400> 276
Met Glu Thr Arg Asp Thr Thr Ala Pro Leu Pro Tyr Ser Tyr Thr Pro
1 5 10 15
Leu Pro Ala Ala Asp Ala Ala Ser Ala Glu Val Thr Gly Thr Gly His
20 25 30
Arg Gly Gly Arg Ser Arg Arg Arg Ser Leu Cys Ala Ala Ala Leu Val
35 40 45
Leu Ser Ala Ala Leu Leu Leu Ala Val Ala Ala Leu Thr Gly Val Gly
50 55 60
Arg Arg Val Asp Val Val Pro Gly Gly Ala Gly Ser Pro Arg Ser Thr
65 70 75 80
Ser Ser Ile Ser Arg Gly Pro Asp Ala Gly Val Ser Glu Lys Thr Ser
85 90 95
Gly Ala Trp Ser Gly Gly Gly Arg Leu Arg Ser Pro Val Tyr Tyr Lys
100 105 110
Gly Trp Tyr His Leu Phe Tyr Gln Tyr Asn Pro Asp Gly Ala Ile Trp
115 120 125
Gly Asn Lys Ile Ala Trp Gly His Ala Val Ser Arg Asp Leu Ile His
130 135 140
Trp Arg His Leu Pro Leu Ala Met Val Pro Asp Gln Trp Tyr Asp Thr
145 150 155 160
Asn Gly Val Trp Thr Gly Ser Ala Thr Thr Leu Pro Asp Gly Arg Leu
165 170 175
Ala Met Leu Tyr Thr Gly Ser Thr Asn Ala Ser Val Gln Val Gln Cys
180 185 190
Leu Ala Val Pro Ala Asp Asp Ala Asp Pro Leu Leu Thr Asn Trp Thr
195 200 205
Lys Tyr Glu Gly Asn Pro Val Leu Tyr Pro Pro Pro Gly Ile Gly Pro
210 215 220
Lys Asp Phe Arg Asp Pro Thr Thr Ala Trp Phe Asp Pro Ser Asp Asn
225 230 235 240
Thr Trp Arg Ile Val Ile Gly Ser Lys Asp Asp Ala Glu Gly Asp His
245 250 255
Ala Gly Ile Ala Val Val Tyr Arg Thr Lys Asp Phe Val Ser Phe Glu
260 265 270
Leu Leu Pro Gly Leu Leu His Arg Val Ala Arg Thr Gly Met Trp Glu
275 280 285
Cys Ile Asp Phe Tyr Pro Val Ala Thr Arg Gly Lys Ala Ser Gly Asn
290 295 300
Gly Val Asp Met Ser Asp Ala Phe Gly Lys Asn Gly Ala Ile Val Gly
305 310 315 320
Asp Val Val His Val Met Lys Ala Ser Met Asp Asp Asp Arg His Asp
325 330 335
Tyr Tyr Ala Leu Gly Arg Tyr Asp Ala Ala Thr Asn Glu Trp Thr Pro
340 345 350
Leu Asp Ala Glu Lys Asp Val Gly Ile Gly Leu Arg Tyr Asp Trp Gly
355 360 365
Lys Phe Tyr Ala Ser Lys Thr Phe Tyr Asp Pro Ala Lys Arg Arg Arg
370 375 380
Val Leu Trp Gly Trp Val Gly Glu Thr Asp Ser Glu Arg Ala Asp Val
385 390 395 400
Ser Lys Gly Trp Ala Ser Leu Gln Gly Ile Pro Arg Thr Val Leu Leu
405 410 415
Asp Thr Lys Thr Gly Ser Asn Leu Leu Gln Trp Pro Val Glu Glu Ala
420 425 430
Glu Thr Leu Arg Thr Asn Ser Thr Asp Leu Ser Gly Ile Thr Ile Asp
435 440 445
Tyr Gly Ser Ala Phe Pro Leu Asn Leu Arg Arg Ala Thr Gln Leu Asp
450 455 460
Ile Glu Ala Glu Phe Gln Leu Asp Arg Arg Ala Val Met Ser Leu Asn
465 470 475 480
Glu Ala Asp Val Gly Tyr Asn Cys Ser Thr Ser Gly Gly Ala Ala Ala
485 490 495
Arg Gly Ala Leu Gly Pro Phe Gly Leu Leu Val Leu Ala Asp Gln His
500 505 510
Leu Arg Glu Gln Thr Ala Val Tyr Phe Tyr Val Ala Lys Gly Leu Asp
515 520 525
Gly Ser Leu Thr Thr His Phe Cys Gln Asp Glu Ser Arg Ser Ser Ser
530 535 540
Ala Asn Asp Ile Val Lys Arg Val Val Gly Ser Ser Val Pro Val Leu
545 550 555 560
Asp Asp Glu Thr Thr Leu Ser Leu Arg Val Leu Val Asp His Ser Ile
565 570 575
Val Glu Ser Phe Ala Gln Gly Gly Arg Ser Thr Ala Thr Ser Arg Val
580 585 590
Tyr Pro Thr Glu Ala Ile Tyr Ala Asn Ala Gly Val Phe Leu Phe Asn
595 600 605
Asn Ala Thr Ala Ala Arg Val Thr Ala Lys Lys Leu Val Val His Glu
610 615 620
Met Asp Ser Ser Tyr Asn His Asp Tyr Met Val Thr Asp Ile
625 630 635
<210> 277
<211> 2022
<212> ДНК
<213> Zea mays
<400> 277
atggagaccc gggacacgga tgcgacgccg ctcccctact cgtacacgcc gctgccggcc
60
gccgacgccg cgtcggccga ggtctccggc accggcagga cgcggagcag gcggcggccc
120
ctctgcgcgg cggcgctcgt gctctccgcc gcgctgctcc tagccgtggc cgcgctcgtc
180
ggcgtcggta gccggcccgg cgcggtgggg atgacagagt cggcggcctc gtcgccgacg
240
ccgagcagga gcaggggccc cgaggccggc gtgtccgaga agacgtccgg cgcgtctgac
300
gacggcggca ggctccgtgg agccggcggg aacgccttcc cgtggagcaa tgcgatgctg
360
cagtggcagc gcacgggatt ccacttccag ccgcagaaga actggatgaa cgaccccaat
420
ggccccgtgt actacaaggg ctggtaccac ctcttctacc agtacaaccc tgacggcgcc
480
atctggggca acaagatcgc gtggggccac gccgtgtccc gcgacctgat ccactggcgc
540
cacctcccgc tggccatggt gcccgaccag tggtacgaca ccaacggcgt gtggacgggg
600
tccgccacca cgctccccga cggccgcctc gccatgctct acacgggctc caccaacgcc
660
tccgtccagg tgcagtgcct ggccgtgccc gccgacgacg ccgacccgct gctcaccaac
720
tggaccaagt acgagggcaa cccggtgctg tacccgcccc cgggcatcgg gcccaaggac
780
ttccgcgacc ccaccacggc ctggatcgac ccctcggacg gcgcatggcg cgtcgtcatc
840
ggctccaagg acgacgacgg ccacgcgggc atcgccgtcg tctaccgcac cacggacctg
900
gtgcacttcg agctcctccc gggcctgctc caccgcgtcg acggcaccgg catgtgggag
960
tgcatcgact tctaccccgt cgccacacga ggcagggcgt cggccaacgg cgtcgacatg
1020
tccgacgcca tcgccagcaa cggcgccgtc gccggggacg tcctgcacgt catgaaggcc
1080
agcatggacg acgaccgcca cgactactac gcgctgggga ggtacgacgc ggccgccaac
1140
gcctggacgc cgatcgacgc cggcagggac gtcggcatcg gcctgcgcta cgactggggc
1200
aagttctacg cgtccaagac gttctacgac ccggccaagc gccgccgcgt gctgtgggga
1260
tgggttggcg agacagactc ggagcgcgcg gacgtgtcca agggatgggc gtcgctgcag
1320
ggtatccccc ggacggtgct cctggacacc aagacgggta gcaacctgct gcagtggccc
1380
gtggaggagg tggagacgct gcgcaccaac tccaccgacc tcagcggcat caccatcgac
1440
tacggctccg tgttcccgct caacctccgc cgcgccaccc agctggacat cgaggcggag
1500
ttccagctgg accgccgcgc cgtcatgtcg ctcaacgagg cggacgtggg ctacaactgc
1560
agcaccagcg ggggcgccgc cggccgcggc gcgctggggc ccttcggcct gctcgtcctc
1620
gccgaccgcc gcctccgccg cgagcagacg gccgtctact tctacgtggc caagggcctg
1680
gacggctccc tcgccacgca cttctgccag gacgagtccc gctcctccag cgccaccgac
1740
atcgtcaagc gcgtcgtcgg cagcgccgtc cccgtgctgg aggacgaggc cacgctctcg
1800
ctccgggtgc tcgtcgacca ctccatcgtc gagagcttcg cgcagggcgg gaggtccacc
1860
gccacatcgc gcgtctaccc caccgaggcc atctacgcca acgccggcgt cttcctcttc
1920
aacaacgcca ccgccgcgcg ggtcacggcc acgaagctcg tcgtccacga gatggactcg
1980
tcatacaacc acgactacat ggcgccggtg gcagacatct ga
2022
<210> 278
<211> 673
<212> БЕЛОК
<213> Zea mays
<400> 278
Met Glu Thr Arg Asp Thr Asp Ala Thr Pro Leu Pro Tyr Ser Tyr Thr
1 5 10 15
Pro Leu Pro Ala Ala Asp Ala Ala Ser Ala Glu Val Ser Gly Thr Gly
20 25 30
Arg Thr Arg Ser Arg Arg Arg Pro Leu Cys Ala Ala Ala Leu Val Leu
35 40 45
Ser Ala Ala Leu Leu Leu Ala Val Ala Ala Leu Val Gly Val Gly Ser
50 55 60
Arg Pro Gly Ala Val Gly Met Thr Glu Ser Ala Ala Ser Ser Pro Thr
65 70 75 80
Pro Ser Arg Ser Arg Gly Pro Glu Ala Gly Val Ser Glu Lys Thr Ser
85 90 95
Gly Ala Ser Asp Asp Gly Gly Arg Leu Arg Gly Ala Gly Gly Asn Ala
100 105 110
Phe Pro Trp Ser Asn Ala Met Leu Gln Trp Gln Arg Thr Gly Phe His
115 120 125
Phe Gln Pro Gln Lys Asn Trp Met Asn Asp Pro Asn Gly Pro Val Tyr
130 135 140
Tyr Lys Gly Trp Tyr His Leu Phe Tyr Gln Tyr Asn Pro Asp Gly Ala
145 150 155 160
Ile Trp Gly Asn Lys Ile Ala Trp Gly His Ala Val Ser Arg Asp Leu
165 170 175
Ile His Trp Arg His Leu Pro Leu Ala Met Val Pro Asp Gln Trp Tyr
180 185 190
Asp Thr Asn Gly Val Trp Thr Gly Ser Ala Thr Thr Leu Pro Asp Gly
195 200 205
Arg Leu Ala Met Leu Tyr Thr Gly Ser Thr Asn Ala Ser Val Gln Val
210 215 220
Gln Cys Leu Ala Val Pro Ala Asp Asp Ala Asp Pro Leu Leu Thr Asn
225 230 235 240
Trp Thr Lys Tyr Glu Gly Asn Pro Val Leu Tyr Pro Pro Pro Gly Ile
245 250 255
Gly Pro Lys Asp Phe Arg Asp Pro Thr Thr Ala Trp Ile Asp Pro Ser
260 265 270
Asp Gly Ala Trp Arg Val Val Ile Gly Ser Lys Asp Asp Asp Gly His
275 280 285
Ala Gly Ile Ala Val Val Tyr Arg Thr Thr Asp Leu Val His Phe Glu
290 295 300
Leu Leu Pro Gly Leu Leu His Arg Val Asp Gly Thr Gly Met Trp Glu
305 310 315 320
Cys Ile Asp Phe Tyr Pro Val Ala Thr Arg Gly Arg Ala Ser Ala Asn
325 330 335
Gly Val Asp Met Ser Asp Ala Ile Ala Ser Asn Gly Ala Val Ala Gly
340 345 350
Asp Val Leu His Val Met Lys Ala Ser Met Asp Asp Asp Arg His Asp
355 360 365
Tyr Tyr Ala Leu Gly Arg Tyr Asp Ala Ala Ala Asn Ala Trp Thr Pro
370 375 380
Ile Asp Ala Gly Arg Asp Val Gly Ile Gly Leu Arg Tyr Asp Trp Gly
385 390 395 400
Lys Phe Tyr Ala Ser Lys Thr Phe Tyr Asp Pro Ala Lys Arg Arg Arg
405 410 415
Val Leu Trp Gly Trp Val Gly Glu Thr Asp Ser Glu Arg Ala Asp Val
420 425 430
Ser Lys Gly Trp Ala Ser Leu Gln Gly Ile Pro Arg Thr Val Leu Leu
435 440 445
Asp Thr Lys Thr Gly Ser Asn Leu Leu Gln Trp Pro Val Glu Glu Val
450 455 460
Glu Thr Leu Arg Thr Asn Ser Thr Asp Leu Ser Gly Ile Thr Ile Asp
465 470 475 480
Tyr Gly Ser Val Phe Pro Leu Asn Leu Arg Arg Ala Thr Gln Leu Asp
485 490 495
Ile Glu Ala Glu Phe Gln Leu Asp Arg Arg Ala Val Met Ser Leu Asn
500 505 510
Glu Ala Asp Val Gly Tyr Asn Cys Ser Thr Ser Gly Gly Ala Ala Gly
515 520 525
Arg Gly Ala Leu Gly Pro Phe Gly Leu Leu Val Leu Ala Asp Arg Arg
530 535 540
Leu Arg Arg Glu Gln Thr Ala Val Tyr Phe Tyr Val Ala Lys Gly Leu
545 550 555 560
Asp Gly Ser Leu Ala Thr His Phe Cys Gln Asp Glu Ser Arg Ser Ser
565 570 575
Ser Ala Thr Asp Ile Val Lys Arg Val Val Gly Ser Ala Val Pro Val
580 585 590
Leu Glu Asp Glu Ala Thr Leu Ser Leu Arg Val Leu Val Asp His Ser
595 600 605
Ile Val Glu Ser Phe Ala Gln Gly Gly Arg Ser Thr Ala Thr Ser Arg
610 615 620
Val Tyr Pro Thr Glu Ala Ile Tyr Ala Asn Ala Gly Val Phe Leu Phe
625 630 635 640
Asn Asn Ala Thr Ala Ala Arg Val Thr Ala Thr Lys Leu Val Val His
645 650 655
Glu Met Asp Ser Ser Tyr Asn His Asp Tyr Met Ala Pro Val Ala Asp
660 665 670
Ile
<210> 279
<211> 2451
<212> ДНК
<213> Sorghum bicolor
<400> 279
atgggggaag ctgccggtga ccgtgttctg agccgcctcc acagcgtcag ggagcgcatt
60
ggcgattcac tctctgccca ccccaatgag cttgtcgccg tcttcaccag gctgaaaaac
120
cttggaaagg gtatgctgca gccccaccag atcattgccg agtacaacag tgctatccct
180
gaggctgaac gtgagaagct gaaggatggt gcctttgagg atgtcctgag ggcagctcag
240
gaagcaattg ttatcccccc atgggttgca cttgccatcc gccctaggcc tggtgtctgg
300
gagtatgtga gggtcaacgt cagcgagctc gctgttgagg agctgagagt ccctgagtac
360
ctgcagttca aggaacagct tgtggaagaa ggccccaaca acaactttgt tcttgagctg
420
gactttgagc cattcaatgc gtccttcccc cgtccttctc tgtcaaagtc cattggcaat
480
ggtgtgcagt tcctcaacag gcacctgtca tcaaagctct tccatgacaa ggagagcatg
540
taccccttgc tcaacttcct tcgtgcccac aactacaagg ggatgaccat gatgttgaac
600
gacagaatcc gcagtctcag tgctctgcaa ggcgctctga ggaaggctga ggagcacctg
660
tccaccctcc aagctgatac cccatactct gaatttcacc acaggttcca ggaacttggt
720
ctggagaagg gttggggtga ctgcgctaag cgcgcacagg agactattca cctcctcttg
780
gaccttcttg aggccccaga tccgtccacc ctggagaagt tccttggaac gatccccatg
840
gtgttcaatg ttgttatcct ctcccctcat ggttactttg ctcaagctaa tgtcttgggt
900
taccctgaca ccggaggcca ggttgtgtac attttggacc aagtccgtgc tatggagaat
960
gaaatgctgc tgaggatcaa gcagtgtggt cttgacatca caccaaagat ccttattgtt
1020
accaggttgc ttcctgatgc aactggcacc acctgtggtc agcgtcttga gaaggtcctt
1080
ggcactgagc actgccatat ccttcgtgtg ccattcagaa cagaaaatgg aattgttcgc
1140
aagtggatct cgcgttttga agtctggcct tacctggaga cttacactga tgatgtggca
1200
catgagattg ctggagagct tcaggccaat cctgacctga tcatcggaaa ctacagtgat
1260
ggaaaccttg tcgcatgttt gctcgcgcac aagatgggtg ttactcactg taccattgcc
1320
cacgcccttg agaaaactaa gtaccctaac tctgacctct actggaagaa gtttgaggac
1380
cactaccact tctcgtgcca gttcaccact gacttgattg ctatgaacca tgctgacttc
1440
attatcacca gtaccttcca agagattgct ggaaacaagg acaccgtcgg tcagtacgag
1500
tcacacatgg cattcacaat gcctggtctg taccgcgttg tccacggtat tgatgtgttt
1560
gaccctaagt tcaacatcgt gtctcctggt gcggacttgt ccatctactt cccatacacc
1620
gagtcacaca agaggttgac ctccctccac ccggagattg aggagctcct gtacagccaa
1680
accgagaaca ccgagcacaa gtttgtgctg aacgacagga acaagccaat catcttctcc
1740
atggctcgtc ttgaccgcgt caagaacttg actggtctgg tggagcttta tggccggaac
1800
aagcgcctgc aggagctggt gaacctcgtg gttgtgtgcg gtgaccacgg caacccgtcc
1860
aaggacaagg aggagcaggc cgagttcaag aagatgtttg acctcatcga gcagtacaac
1920
ctgaacgggc acatccgctg gatctccgcc cagatgaacc gtgtccgcaa cggtgagctg
1980
taccgctaca tttgcgacac caagggtgcc ttcgtgcagc ctgctttcta cgaggcgttc
2040
gggctgacgg tggttgaggc catgacctgt ggcctgccca cgttcgccac cgcctatggt
2100
ggtccggctg agatcatcgt gcacggcgtg tctggcttcc acattgaccc gtaccagggc
2160
gacaaggcgt cggcgctgct cgtggacttc tttgagaagt gccagacgga ttcgagccac
2220
tggaacaaaa tctcccaggg cgggctccag cgtatcgagg agaaatacac ctggaagctg
2280
tactcggaga ggctgatgac cctgacgggc gtgtatggtt tctggaagta cgtgtccaac
2340
ctggagaggc gcgagacccg gcggtacctg gagatgctgt acgcgctcaa gtaccgcacc
2400
atggccagca ccgtgccgtt ggccgtggag ggagagccct ccagcaagtg a
2451
<210> 280
<211> 816
<212> БЕЛОК
<213> Sorghum bicolor
<400> 280
Met Gly Glu Ala Ala Gly Asp Arg Val Leu Ser Arg Leu His Ser Val
1 5 10 15
Arg Glu Arg Ile Gly Asp Ser Leu Ser Ala His Pro Asn Glu Leu Val
20 25 30
Ala Val Phe Thr Arg Leu Lys Asn Leu Gly Lys Gly Met Leu Gln Pro
35 40 45
His Gln Ile Ile Ala Glu Tyr Asn Ser Ala Ile Pro Glu Ala Glu Arg
50 55 60
Glu Lys Leu Lys Asp Gly Ala Phe Glu Asp Val Leu Arg Ala Ala Gln
65 70 75 80
Glu Ala Ile Val Ile Pro Pro Trp Val Ala Leu Ala Ile Arg Pro Arg
85 90 95
Pro Gly Val Trp Glu Tyr Val Arg Val Asn Val Ser Glu Leu Ala Val
100 105 110
Glu Glu Leu Arg Val Pro Glu Tyr Leu Gln Phe Lys Glu Gln Leu Val
115 120 125
Glu Glu Gly Pro Asn Asn Asn Phe Val Leu Glu Leu Asp Phe Glu Pro
130 135 140
Phe Asn Ala Ser Phe Pro Arg Pro Ser Leu Ser Lys Ser Ile Gly Asn
145 150 155 160
Gly Val Gln Phe Leu Asn Arg His Leu Ser Ser Lys Leu Phe His Asp
165 170 175
Lys Glu Ser Met Tyr Pro Leu Leu Asn Phe Leu Arg Ala His Asn Tyr
180 185 190
Lys Gly Met Thr Met Met Leu Asn Asp Arg Ile Arg Ser Leu Ser Ala
195 200 205
Leu Gln Gly Ala Leu Arg Lys Ala Glu Glu His Leu Ser Thr Leu Gln
210 215 220
Ala Asp Thr Pro Tyr Ser Glu Phe His His Arg Phe Gln Glu Leu Gly
225 230 235 240
Leu Glu Lys Gly Trp Gly Asp Cys Ala Lys Arg Ala Gln Glu Thr Ile
245 250 255
His Leu Leu Leu Asp Leu Leu Glu Ala Pro Asp Pro Ser Thr Leu Glu
260 265 270
Lys Phe Leu Gly Thr Ile Pro Met Val Phe Asn Val Val Ile Leu Ser
275 280 285
Pro His Gly Tyr Phe Ala Gln Ala Asn Val Leu Gly Tyr Pro Asp Thr
290 295 300
Gly Gly Gln Val Val Tyr Ile Leu Asp Gln Val Arg Ala Met Glu Asn
305 310 315 320
Glu Met Leu Leu Arg Ile Lys Gln Cys Gly Leu Asp Ile Thr Pro Lys
325 330 335
Ile Leu Ile Val Thr Arg Leu Leu Pro Asp Ala Thr Gly Thr Thr Cys
340 345 350
Gly Gln Arg Leu Glu Lys Val Leu Gly Thr Glu His Cys His Ile Leu
355 360 365
Arg Val Pro Phe Arg Thr Glu Asn Gly Ile Val Arg Lys Trp Ile Ser
370 375 380
Arg Phe Glu Val Trp Pro Tyr Leu Glu Thr Tyr Thr Asp Asp Val Ala
385 390 395 400
His Glu Ile Ala Gly Glu Leu Gln Ala Asn Pro Asp Leu Ile Ile Gly
405 410 415
Asn Tyr Ser Asp Gly Asn Leu Val Ala Cys Leu Leu Ala His Lys Met
420 425 430
Gly Val Thr His Cys Thr Ile Ala His Ala Leu Glu Lys Thr Lys Tyr
435 440 445
Pro Asn Ser Asp Leu Tyr Trp Lys Lys Phe Glu Asp His Tyr His Phe
450 455 460
Ser Cys Gln Phe Thr Thr Asp Leu Ile Ala Met Asn His Ala Asp Phe
465 470 475 480
Ile Ile Thr Ser Thr Phe Gln Glu Ile Ala Gly Asn Lys Asp Thr Val
485 490 495
Gly Gln Tyr Glu Ser His Met Ala Phe Thr Met Pro Gly Leu Tyr Arg
500 505 510
Val Val His Gly Ile Asp Val Phe Asp Pro Lys Phe Asn Ile Val Ser
515 520 525
Pro Gly Ala Asp Leu Ser Ile Tyr Phe Pro Tyr Thr Glu Ser His Lys
530 535 540
Arg Leu Thr Ser Leu His Pro Glu Ile Glu Glu Leu Leu Tyr Ser Gln
545 550 555 560
Thr Glu Asn Thr Glu His Lys Phe Val Leu Asn Asp Arg Asn Lys Pro
565 570 575
Ile Ile Phe Ser Met Ala Arg Leu Asp Arg Val Lys Asn Leu Thr Gly
580 585 590
Leu Val Glu Leu Tyr Gly Arg Asn Lys Arg Leu Gln Glu Leu Val Asn
595 600 605
Leu Val Val Val Cys Gly Asp His Gly Asn Pro Ser Lys Asp Lys Glu
610 615 620
Glu Gln Ala Glu Phe Lys Lys Met Phe Asp Leu Ile Glu Gln Tyr Asn
625 630 635 640
Leu Asn Gly His Ile Arg Trp Ile Ser Ala Gln Met Asn Arg Val Arg
645 650 655
Asn Gly Glu Leu Tyr Arg Tyr Ile Cys Asp Thr Lys Gly Ala Phe Val
660 665 670
Gln Pro Ala Phe Tyr Glu Ala Phe Gly Leu Thr Val Val Glu Ala Met
675 680 685
Thr Cys Gly Leu Pro Thr Phe Ala Thr Ala Tyr Gly Gly Pro Ala Glu
690 695 700
Ile Ile Val His Gly Val Ser Gly Phe His Ile Asp Pro Tyr Gln Gly
705 710 715 720
Asp Lys Ala Ser Ala Leu Leu Val Asp Phe Phe Glu Lys Cys Gln Thr
725 730 735
Asp Ser Ser His Trp Asn Lys Ile Ser Gln Gly Gly Leu Gln Arg Ile
740 745 750
Glu Glu Lys Tyr Thr Trp Lys Leu Tyr Ser Glu Arg Leu Met Thr Leu
755 760 765
Thr Gly Val Tyr Gly Phe Trp Lys Tyr Val Ser Asn Leu Glu Arg Arg
770 775 780
Glu Thr Arg Arg Tyr Leu Glu Met Leu Tyr Ala Leu Lys Tyr Arg Thr
785 790 795 800
Met Ala Ser Thr Val Pro Leu Ala Val Glu Gly Glu Pro Ser Ser Lys
805 810 815
<210> 281
<211> 2451
<212> ДНК
<213> Zea mays
<400> 281
atgggggaag gtgcaggtga ccgtgtcctg agccgcctcc acagcgtcag ggagcgcatt
60
ggcgactcac tctctgccca ccccaatgag cttgtcgccg tcttcaccag gctgaaaaac
120
cttggaaagg gtatgctgca gccccaccag atcattgccg agtacaacaa tgcgatccct
180
gaggctgagc gcgagaagct caaggatggt gcttttgagg atgtcctgag ggcagctcag
240
gaggcgattg tcatcccccc atgggttgca cttgccatcc gccctaggcc tggtgtctgg
300
gagtatgtga gggtcaacgt cagtgagctc gctgttgagg agctgagagt tcctgagtac
360
ctgcagttca aggaacagct tgtggaagaa ggccccaaca acaactttgt tcttgagctg
420
gactttgagc cattcaatgc ctccttcccc cgtccttctc tgtcaaagtc cattggcaat
480
ggcgtgcagt tcctcaacag gcacctgtca tcaaagctct tccatgacaa ggagagcatg
540
taccccttgc tcaacttcct tcgcgcccac aactacaagg ggatgaccat gatgttgaac
600
gacagaatcc gcagtctcag tgctctgcaa ggtgcgctga ggaaggctga ggagcacctg
660
tccaccctac aagctgatac cccatactct gaatttcacc acaggttcca ggaacttggt
720
ctggagaagg gttggggtga ttgcgctaag cgtgcacagg agactatcca cctcctcttg
780
gacctcctgg aggccccaga tccgtccacc ctggagaagt tccttggaac gatccccatg
840
gtgttcaatg tcgttatcct ctcccctcat ggttacttcg ctcaagctaa tgtcttgggt
900
taccctgaca ccggaggcca ggttgtctac atcttggatc aagtgcgcgc tatggagaac
960
gaaatgctgc tgaggatcaa gcagtgtggt cttgacatca cgccgaagat ccttattgtc
1020
accaggttgc tccctgatgc aactggcacc acctgtggcc agcgccttga gaaggtcctt
1080
ggcaccgagc actgccatat ccttcgcgtg ccattcagaa cagaaaacgg aatcgttcgc
1140
aagtggatct cgcgatttga agtctggccg tacctggaga cttacactga tgacgtggcg
1200
catgagattg ctggagagct tcaggccaat cctgacctga tcatcggaaa ctacagtgac
1260
ggaaaccttg ttgcgtgttt gctcgcccac aagatgggtg ttactcactg taccattgcc
1320
catgcgcttg agaaaactaa gtaccctaac tccgacctct actggaagaa gtttgaggat
1380
cactaccact tctcgtgcca gttcaccact gacttgattg caatgaacca tgccgacttc
1440
atcatcacca gtaccttcca agagatcgcc ggaaacaagg acaccgtcgg ccagtacgag
1500
tcacacatgg cgttcacaat gcctggcctg taccgcgttg tccacggcat tgatgtgttc
1560
gaccccaagt tcaacatcgt gtctcctggc gcggacctgt ccatctactt cccgtacacc
1620
gagtcgcaca agaggctgac ctcccttcac ccggagattg aggagctcct gtacagccaa
1680
accgagaaca cggagcacaa gttcgttctg aacgacagga acaagccaat catcttctcc
1740
atggctcgtc tcgaccgtgt gaagaacttg actgggctgg tggagctgta cggccggaac
1800
aagcggctgc aggagctggt gaacctcgtg gtcgtctgcg gcgaccatgg caacccttcc
1860
aaggacaagg aggagcaggc cgagttcaag aagatgtttg acctcatcga gcagtacaac
1920
ctgaacgggc acatccgctg gatctccgcc cagatgaacc gcgtccgcaa cggcgagctg
1980
taccgctaca tctgcgacac caagggcgcc ttcgtgcagc ctgctttcta cgaggctttc
2040
gggctgacgg tggttgaggc catgacctgc ggcctgccca cgttcgccac cgcctacggc
2100
ggtccggccg agatcatcgt gcacggcgtg tctggctacc acatcgaccc ttaccagggc
2160
gacaaggcgt cggccctgct cgtggacttc ttcgacaagt gccaggcgga gccgagccac
2220
tggagcaaga tctcccaggg cgggctccag cgtatcgagg agaagtacac ctggaagctg
2280
tactcggaga ggctgatgac cctcaccggc gtgtacgggt tctggaagta cgtgtccaac
2340
ctggagaggc gcgagacccg gcggtacctg gagatgctgt acgcgctcaa gtaccgcacc
2400
atggcgagca ccgtgccgct ggccgtggag ggagagccct ccagcaagtg a
2451
<210> 282
<211> 816
<212> БЕЛОК
<213> Zea mays
<400> 282
Met Gly Glu Gly Ala Gly Asp Arg Val Leu Ser Arg Leu His Ser Val
1 5 10 15
Arg Glu Arg Ile Gly Asp Ser Leu Ser Ala His Pro Asn Glu Leu Val
20 25 30
Ala Val Phe Thr Arg Leu Lys Asn Leu Gly Lys Gly Met Leu Gln Pro
35 40 45
His Gln Ile Ile Ala Glu Tyr Asn Asn Ala Ile Pro Glu Ala Glu Arg
50 55 60
Glu Lys Leu Lys Asp Gly Ala Phe Glu Asp Val Leu Arg Ala Ala Gln
65 70 75 80
Glu Ala Ile Val Ile Pro Pro Trp Val Ala Leu Ala Ile Arg Pro Arg
85 90 95
Pro Gly Val Trp Glu Tyr Val Arg Val Asn Val Ser Glu Leu Ala Val
100 105 110
Glu Glu Leu Arg Val Pro Glu Tyr Leu Gln Phe Lys Glu Gln Leu Val
115 120 125
Glu Glu Gly Pro Asn Asn Asn Phe Val Leu Glu Leu Asp Phe Glu Pro
130 135 140
Phe Asn Ala Ser Phe Pro Arg Pro Ser Leu Ser Lys Ser Ile Gly Asn
145 150 155 160
Gly Val Gln Phe Leu Asn Arg His Leu Ser Ser Lys Leu Phe His Asp
165 170 175
Lys Glu Ser Met Tyr Pro Leu Leu Asn Phe Leu Arg Ala His Asn Tyr
180 185 190
Lys Gly Met Thr Met Met Leu Asn Asp Arg Ile Arg Ser Leu Ser Ala
195 200 205
Leu Gln Gly Ala Leu Arg Lys Ala Glu Glu His Leu Ser Thr Leu Gln
210 215 220
Ala Asp Thr Pro Tyr Ser Glu Phe His His Arg Phe Gln Glu Leu Gly
225 230 235 240
Leu Glu Lys Gly Trp Gly Asp Cys Ala Lys Arg Ala Gln Glu Thr Ile
245 250 255
His Leu Leu Leu Asp Leu Leu Glu Ala Pro Asp Pro Ser Thr Leu Glu
260 265 270
Lys Phe Leu Gly Thr Ile Pro Met Val Phe Asn Val Val Ile Leu Ser
275 280 285
Pro His Gly Tyr Phe Ala Gln Ala Asn Val Leu Gly Tyr Pro Asp Thr
290 295 300
Gly Gly Gln Val Val Tyr Ile Leu Asp Gln Val Arg Ala Met Glu Asn
305 310 315 320
Glu Met Leu Leu Arg Ile Lys Gln Cys Gly Leu Asp Ile Thr Pro Lys
325 330 335
Ile Leu Ile Val Thr Arg Leu Leu Pro Asp Ala Thr Gly Thr Thr Cys
340 345 350
Gly Gln Arg Leu Glu Lys Val Leu Gly Thr Glu His Cys His Ile Leu
355 360 365
Arg Val Pro Phe Arg Thr Glu Asn Gly Ile Val Arg Lys Trp Ile Ser
370 375 380
Arg Phe Glu Val Trp Pro Tyr Leu Glu Thr Tyr Thr Asp Asp Val Ala
385 390 395 400
His Glu Ile Ala Gly Glu Leu Gln Ala Asn Pro Asp Leu Ile Ile Gly
405 410 415
Asn Tyr Ser Asp Gly Asn Leu Val Ala Cys Leu Leu Ala His Lys Met
420 425 430
Gly Val Thr His Cys Thr Ile Ala His Ala Leu Glu Lys Thr Lys Tyr
435 440 445
Pro Asn Ser Asp Leu Tyr Trp Lys Lys Phe Glu Asp His Tyr His Phe
450 455 460
Ser Cys Gln Phe Thr Thr Asp Leu Ile Ala Met Asn His Ala Asp Phe
465 470 475 480
Ile Ile Thr Ser Thr Phe Gln Glu Ile Ala Gly Asn Lys Asp Thr Val
485 490 495
Gly Gln Tyr Glu Ser His Met Ala Phe Thr Met Pro Gly Leu Tyr Arg
500 505 510
Val Val His Gly Ile Asp Val Phe Asp Pro Lys Phe Asn Ile Val Ser
515 520 525
Pro Gly Ala Asp Leu Ser Ile Tyr Phe Pro Tyr Thr Glu Ser His Lys
530 535 540
Arg Leu Thr Ser Leu His Pro Glu Ile Glu Glu Leu Leu Tyr Ser Gln
545 550 555 560
Thr Glu Asn Thr Glu His Lys Phe Val Leu Asn Asp Arg Asn Lys Pro
565 570 575
Ile Ile Phe Ser Met Ala Arg Leu Asp Arg Val Lys Asn Leu Thr Gly
580 585 590
Leu Val Glu Leu Tyr Gly Arg Asn Lys Arg Leu Gln Glu Leu Val Asn
595 600 605
Leu Val Val Val Cys Gly Asp His Gly Asn Pro Ser Lys Asp Lys Glu
610 615 620
Glu Gln Ala Glu Phe Lys Lys Met Phe Asp Leu Ile Glu Gln Tyr Asn
625 630 635 640
Leu Asn Gly His Ile Arg Trp Ile Ser Ala Gln Met Asn Arg Val Arg
645 650 655
Asn Gly Glu Leu Tyr Arg Tyr Ile Cys Asp Thr Lys Gly Ala Phe Val
660 665 670
Gln Pro Ala Phe Tyr Glu Ala Phe Gly Leu Thr Val Val Glu Ala Met
675 680 685
Thr Cys Gly Leu Pro Thr Phe Ala Thr Ala Tyr Gly Gly Pro Ala Glu
690 695 700
Ile Ile Val His Gly Val Ser Gly Tyr His Ile Asp Pro Tyr Gln Gly
705 710 715 720
Asp Lys Ala Ser Ala Leu Leu Val Asp Phe Phe Asp Lys Cys Gln Ala
725 730 735
Glu Pro Ser His Trp Ser Lys Ile Ser Gln Gly Gly Leu Gln Arg Ile
740 745 750
Glu Glu Lys Tyr Thr Trp Lys Leu Tyr Ser Glu Arg Leu Met Thr Leu
755 760 765
Thr Gly Val Tyr Gly Phe Trp Lys Tyr Val Ser Asn Leu Glu Arg Arg
770 775 780
Glu Thr Arg Arg Tyr Leu Glu Met Leu Tyr Ala Leu Lys Tyr Arg Thr
785 790 795 800
Met Ala Ser Thr Val Pro Leu Ala Val Glu Gly Glu Pro Ser Ser Lys
805 810 815
<210> 283
<211> 1680
<212> ДНК
<213> Sorghum bicolor
<400> 283
atggagctgg ctgtcggcgg cgggatgcgg cggtcggcgt cgcacacctc gctgtcggag
60
tcggacgact tcgagctcac gcggctgctc agcaagccgc ggatcaacgt cgagcgccag
120
cgctccttcg acgaccgctc gctcagcgac gtctcgcact cgggcggcta cggcagggga
180
ggcttcgacg gcatgtactc gcctgggggc ggcctgcgct ccctcgtcgg cacgccggca
240
tcctccgcgc tgcactcctt cgagccccac cccatcgtcg gcgacgcatg ggaggcgctc
300
cgccgctcac tcgtcttctt tcgcggccag ccccttggca ccgtcgccgc cgtcgaccac
360
gcttccgagg aagtcctcaa ctacgaccaa gtgtttgtga gggatttcgt gccgagcgcg
420
ctggcgtttc tgatgaatgg cgagccggat atcgtcaaga acttccttct gaagaccctg
480
ctgctgcagg gctgggagaa gaaagtcgac cggttcaagc ttggggaggg agccatgccg
540
gctagcttca aggtgatgca tgacgccaag aagggggtcg agaccctaca tgctgatttt
600
ggagagagcg ccattgggag ggttgcgcct gtggattctg gtttctggtg gatcatactc
660
ctccgggcgt acacaaaaac caccggtgat atgaccctgg cagagacacc ggagtgccag
720
aaagggatga ggctcatact cagcctgtgc ttatctgagg ggtttgatac cttcccgaca
780
ttgttatgtg ctgatggttg ctgtatgata gatcgcagaa tgggtgtata tggataccct
840
attgagattc aagccctttt ctttatggca ctaaggtgtg ctcttcaaat gcttaagcat
900
gataatgaag ggaaggaatt tgtagagaag attgctaccc gccttcatgc tttaagttat
960
cacatgcgaa gttatttttg gctcgatttc caacagctaa atgacattta tcgttacaag
1020
acagaagaat attcccacac agccgtcaac aaattcaatg ttattcctga ttcaattccg
1080
gactggctat ttgactttat gccttgtcag ggtgggtttt tcattggtaa tgttagtcct
1140
gccaggatgg acttccgatg gtttgcactt ggaaacatga ttgctatact ttcttctctt
1200
gcaacacctg agcaatctgt tgctataatg gatcttattg aggagcgttg ggaagagctt
1260
atcggtgata tgcctctgaa gatatgttat cctgctattg agaaccatga atggcgaatt
1320
gtgacaggct gtgatccaaa aaatactaga tggagctatc acaatggagg atcgtggcca
1380
gtacttctgt ggctgctgac tgcagcctgc atcaaaactg gacggccgca aattgcaaga
1440
agagcaattg acctagcaga gaggaggctg ttgaaggatg gctggcctga gtactatgac
1500
gggaagcttg gtcggtatgt tggcaagcag gcaaggaaat tccagacttg gtccatcgcg
1560
gggtatttgg tcgccaagat gatgttggaa gatccttcac atcttggcat gatctcccta
1620
gaagaggata aggcgatgtt gaagcctgtt ttgaagcggt ccgcatcatg gacaaactaa
1680
<210> 284
<211> 559
<212> БЕЛОК
<213> Sorghum bicolor
<400> 284
Met Glu Leu Ala Val Gly Gly Gly Met Arg Arg Ser Ala Ser His Thr
1 5 10 15
Ser Leu Ser Glu Ser Asp Asp Phe Glu Leu Thr Arg Leu Leu Ser Lys
20 25 30
Pro Arg Ile Asn Val Glu Arg Gln Arg Ser Phe Asp Asp Arg Ser Leu
35 40 45
Ser Asp Val Ser His Ser Gly Gly Tyr Gly Arg Gly Gly Phe Asp Gly
50 55 60
Met Tyr Ser Pro Gly Gly Gly Leu Arg Ser Leu Val Gly Thr Pro Ala
65 70 75 80
Ser Ser Ala Leu His Ser Phe Glu Pro His Pro Ile Val Gly Asp Ala
85 90 95
Trp Glu Ala Leu Arg Arg Ser Leu Val Phe Phe Arg Gly Gln Pro Leu
100 105 110
Gly Thr Val Ala Ala Val Asp His Ala Ser Glu Glu Val Leu Asn Tyr
115 120 125
Asp Gln Val Phe Val Arg Asp Phe Val Pro Ser Ala Leu Ala Phe Leu
130 135 140
Met Asn Gly Glu Pro Asp Ile Val Lys Asn Phe Leu Leu Lys Thr Leu
145 150 155 160
Leu Leu Gln Gly Trp Glu Lys Lys Val Asp Arg Phe Lys Leu Gly Glu
165 170 175
Gly Ala Met Pro Ala Ser Phe Lys Val Met His Asp Ala Lys Lys Gly
180 185 190
Val Glu Thr Leu His Ala Asp Phe Gly Glu Ser Ala Ile Gly Arg Val
195 200 205
Ala Pro Val Asp Ser Gly Phe Trp Trp Ile Ile Leu Leu Arg Ala Tyr
210 215 220
Thr Lys Thr Thr Gly Asp Met Thr Leu Ala Glu Thr Pro Glu Cys Gln
225 230 235 240
Lys Gly Met Arg Leu Ile Leu Ser Leu Cys Leu Ser Glu Gly Phe Asp
245 250 255
Thr Phe Pro Thr Leu Leu Cys Ala Asp Gly Cys Cys Met Ile Asp Arg
260 265 270
Arg Met Gly Val Tyr Gly Tyr Pro Ile Glu Ile Gln Ala Leu Phe Phe
275 280 285
Met Ala Leu Arg Cys Ala Leu Gln Met Leu Lys His Asp Asn Glu Gly
290 295 300
Lys Glu Phe Val Glu Lys Ile Ala Thr Arg Leu His Ala Leu Ser Tyr
305 310 315 320
His Met Arg Ser Tyr Phe Trp Leu Asp Phe Gln Gln Leu Asn Asp Ile
325 330 335
Tyr Arg Tyr Lys Thr Glu Glu Tyr Ser His Thr Ala Val Asn Lys Phe
340 345 350
Asn Val Ile Pro Asp Ser Ile Pro Asp Trp Leu Phe Asp Phe Met Pro
355 360 365
Cys Gln Gly Gly Phe Phe Ile Gly Asn Val Ser Pro Ala Arg Met Asp
370 375 380
Phe Arg Trp Phe Ala Leu Gly Asn Met Ile Ala Ile Leu Ser Ser Leu
385 390 395 400
Ala Thr Pro Glu Gln Ser Val Ala Ile Met Asp Leu Ile Glu Glu Arg
405 410 415
Trp Glu Glu Leu Ile Gly Asp Met Pro Leu Lys Ile Cys Tyr Pro Ala
420 425 430
Ile Glu Asn His Glu Trp Arg Ile Val Thr Gly Cys Asp Pro Lys Asn
435 440 445
Thr Arg Trp Ser Tyr His Asn Gly Gly Ser Trp Pro Val Leu Leu Trp
450 455 460
Leu Leu Thr Ala Ala Cys Ile Lys Thr Gly Arg Pro Gln Ile Ala Arg
465 470 475 480
Arg Ala Ile Asp Leu Ala Glu Arg Arg Leu Leu Lys Asp Gly Trp Pro
485 490 495
Glu Tyr Tyr Asp Gly Lys Leu Gly Arg Tyr Val Gly Lys Gln Ala Arg
500 505 510
Lys Phe Gln Thr Trp Ser Ile Ala Gly Tyr Leu Val Ala Lys Met Met
515 520 525
Leu Glu Asp Pro Ser His Leu Gly Met Ile Ser Leu Glu Glu Asp Lys
530 535 540
Ala Met Leu Lys Pro Val Leu Lys Arg Ser Ala Ser Trp Thr Asn
545 550 555
<210> 285
<211> 1680
<212> ДНК
<213> Zea mays
<400> 285
atggagctgg ctgtcggcgg cgggatgcgg cggtcggcgt cgcacacctc gctgtcggag
60
tcggatgact tcgagctcac gcggctgctc agcaagccgc ggatcaacgt cgagcgccag
120
cgctccttcg acgaccgctc gcttagcgac gtctcgcact cgggcgggta cggcagggga
180
ggcttcgacg gcatgtactc gcctgggggc ggcctgcgct ccctcgtcgg cacgccggcc
240
tcctccgggc tgcactcctt cgagccccac cccatcgtcg gcgacgcctg ggaggcgctc
300
cgccgctccc tcgtcttgtt ccgcggccag ccccttggca ccgttgccgc cgtcgaccac
360
gcgtccgagg aagtcctcaa ctacgaccaa gtgtttgtga gggatttcgt gccgagcgcg
420
ctggcgtttc tgatgaatgg cgagccagat atcgtcaaga acttccttct gaagaccctg
480
ctgctgcagg gctgggagaa gaaagtcgac cggttcaagc tcggggaggg tgccatgccg
540
gctagcttca aggttatgca tgacgccaag aagggggtcg agaccctaca tgctgatttt
600
ggggagagcg ccattgggag ggttgcgcct gtggattcgg ggttctggtg gatcatactc
660
ctccgggcct acacaaaaac caccggtgat ttgacgctgg cagagacgcc ggagtgccag
720
aaagggatga ggctcatact cagcctgtgc ttatccgagg ggttcgatac cttcccgaca
780
ttgttatgtg ctgatggttg ctgtatgata gatcgcagaa tgggtgtata tggctaccct
840
attgagattc aggccctttt ctttatggca ctaaggtgtg ctcttcaaat gcttaagcat
900
gataatgaag ggaaggagtt tgtagagaag attgctactc gccttcacgc tttaagttat
960
cacatgcgga gttatttttg gctcgatttc caacagctaa atgacatcta tcgttacaag
1020
acagaagaat attcccacac tgctgtcaac aaattcaatg tcattcctga ttcaattccg
1080
gattggctat ttgactttat gccttgtcag ggtgggtttt tcattggtaa tgtcagtcct
1140
gccaggatgg acttccgatg gttcgcactt ggaaacatga ttgctatact ttcttccctt
1200
gcaacacctg agcaatctgt tgctataatg gatcttattg aggagcgttg ggaagagctc
1260
attggtgaaa tgcctctgaa gatatgttat cctgctattg agaaccatga atggcgaatt
1320
gtgacgggct gtgatccaaa aaacactaga tggagttatc ataatggagg atcgtggcca
1380
gtacttctgt ggctgctgac tgcagcctgc atcaaaactg gacggccaca aatcgcaaga
1440
agggcaattg acctagcaga aaggaggctg ttgaaggatg gatggcctga gtactatgat
1500
gggaagcttg gtcggtatgt tggcaagcag gcgaggaaat tccagacctg gtccatcgcg
1560
gggtatttgg tcgccaagat gatgttggaa gatccttcac atctcggcat gatctccctg
1620
gaagaggata gggcaatgtt gaagcctgtt ttgaagcggt ctgcgtcatg gacaaactga
1680
<210> 286
<211> 559
<212> БЕЛОК
<213> Zea mays
<400> 286
Met Glu Leu Ala Val Gly Gly Gly Met Arg Arg Ser Ala Ser His Thr
1 5 10 15
Ser Leu Ser Glu Ser Asp Asp Phe Glu Leu Thr Arg Leu Leu Ser Lys
20 25 30
Pro Arg Ile Asn Val Glu Arg Gln Arg Ser Phe Asp Asp Arg Ser Leu
35 40 45
Ser Asp Val Ser His Ser Gly Gly Tyr Gly Arg Gly Gly Phe Asp Gly
50 55 60
Met Tyr Ser Pro Gly Gly Gly Leu Arg Ser Leu Val Gly Thr Pro Ala
65 70 75 80
Ser Ser Gly Leu His Ser Phe Glu Pro His Pro Ile Val Gly Asp Ala
85 90 95
Trp Glu Ala Leu Arg Arg Ser Leu Val Leu Phe Arg Gly Gln Pro Leu
100 105 110
Gly Thr Val Ala Ala Val Asp His Ala Ser Glu Glu Val Leu Asn Tyr
115 120 125
Asp Gln Val Phe Val Arg Asp Phe Val Pro Ser Ala Leu Ala Phe Leu
130 135 140
Met Asn Gly Glu Pro Asp Ile Val Lys Asn Phe Leu Leu Lys Thr Leu
145 150 155 160
Leu Leu Gln Gly Trp Glu Lys Lys Val Asp Arg Phe Lys Leu Gly Glu
165 170 175
Gly Ala Met Pro Ala Ser Phe Lys Val Met His Asp Ala Lys Lys Gly
180 185 190
Val Glu Thr Leu His Ala Asp Phe Gly Glu Ser Ala Ile Gly Arg Val
195 200 205
Ala Pro Val Asp Ser Gly Phe Trp Trp Ile Ile Leu Leu Arg Ala Tyr
210 215 220
Thr Lys Thr Thr Gly Asp Leu Thr Leu Ala Glu Thr Pro Glu Cys Gln
225 230 235 240
Lys Gly Met Arg Leu Ile Leu Ser Leu Cys Leu Ser Glu Gly Phe Asp
245 250 255
Thr Phe Pro Thr Leu Leu Cys Ala Asp Gly Cys Cys Met Ile Asp Arg
260 265 270
Arg Met Gly Val Tyr Gly Tyr Pro Ile Glu Ile Gln Ala Leu Phe Phe
275 280 285
Met Ala Leu Arg Cys Ala Leu Gln Met Leu Lys His Asp Asn Glu Gly
290 295 300
Lys Glu Phe Val Glu Lys Ile Ala Thr Arg Leu His Ala Leu Ser Tyr
305 310 315 320
His Met Arg Ser Tyr Phe Trp Leu Asp Phe Gln Gln Leu Asn Asp Ile
325 330 335
Tyr Arg Tyr Lys Thr Glu Glu Tyr Ser His Thr Ala Val Asn Lys Phe
340 345 350
Asn Val Ile Pro Asp Ser Ile Pro Asp Trp Leu Phe Asp Phe Met Pro
355 360 365
Cys Gln Gly Gly Phe Phe Ile Gly Asn Val Ser Pro Ala Arg Met Asp
370 375 380
Phe Arg Trp Phe Ala Leu Gly Asn Met Ile Ala Ile Leu Ser Ser Leu
385 390 395 400
Ala Thr Pro Glu Gln Ser Val Ala Ile Met Asp Leu Ile Glu Glu Arg
405 410 415
Trp Glu Glu Leu Ile Gly Glu Met Pro Leu Lys Ile Cys Tyr Pro Ala
420 425 430
Ile Glu Asn His Glu Trp Arg Ile Val Thr Gly Cys Asp Pro Lys Asn
435 440 445
Thr Arg Trp Ser Tyr His Asn Gly Gly Ser Trp Pro Val Leu Leu Trp
450 455 460
Leu Leu Thr Ala Ala Cys Ile Lys Thr Gly Arg Pro Gln Ile Ala Arg
465 470 475 480
Arg Ala Ile Asp Leu Ala Glu Arg Arg Leu Leu Lys Asp Gly Trp Pro
485 490 495
Glu Tyr Tyr Asp Gly Lys Leu Gly Arg Tyr Val Gly Lys Gln Ala Arg
500 505 510
Lys Phe Gln Thr Trp Ser Ile Ala Gly Tyr Leu Val Ala Lys Met Met
515 520 525
Leu Glu Asp Pro Ser His Leu Gly Met Ile Ser Leu Glu Glu Asp Arg
530 535 540
Ala Met Leu Lys Pro Val Leu Lys Arg Ser Ala Ser Trp Thr Asn
545 550 555
<210> 287
<211> 1785
<212> ДНК
<213> Sorghum bicolor
<400> 287
atggacgccg gcaccggggg cggcgggcca acggccatcc gcgtgcccta ccgccacctc
60
cgcgacgccg agatggagct cgtcagcctc aacggagcgg acgcgggccc gacgccgcac
120
aaggacgcgg accagccgcg gagccgcggc gccaacgccg acaggaccaa gctcgtgctt
180
gcctgcatgg tcgccgcggg cgtccagttc gggtgggcac tgcagctctc cctcctcacg
240
ccatacatcc agaccctagg aatagaccat gccatggcat catttatttg gctctgtggg
300
ccaataactg gttttgtggt tcaaccttgt gttggtgttt ggagtgataa gtgccgatca
360
aagtatggga ggagaaggcc atttattttg gctggatgca taatgatatg tgctgctgta
420
actttaatcg ggttttctgc agacctcggt tacatcttag gggataccac cgagcactgc
480
agaacatata aaggttcaag atttcgagct gctatggttt tcattctagg attctggatg
540
ttggacctgg caaacaatac agtgcaaggt cctgctcgtg ctcttctagc tgatctttca
600
ggtcctgatc agtgtaattc tgcaaatgca atattctgct catggatggc tgttggaaat
660
attcttggtt tttcagctgg tgcaagcggg gattggcaca agtggtttcc ttttctaatg
720
actagagcct gctgtgaagc ttgtggtaat ttgaaagcag ctttcttagt tgcagtcgta
780
tttcttttgt tctgtatgtc tgttacgctg tacttcgctg aagagatccc actagagcca
840
aaagatgcac aaggactgtc agattctgct cctctactga acggttctag agaggatgca
900
catgcattga atgaaccaaa taatgaaaga tttcctaatg gccatgtaga tggaaacaac
960
gtgtcggcta acaacaacac tgaggaattt ccaaatgcga attccaacac agacaatgga
1020
ggagtcttca atgatggacc tggagcagtt ttggttaaca ttttgaccag catgaggcat
1080
ctacctcctg ggatgcattc agtgcttgta gttatggccc taacatggtt gtcatggttt
1140
cccttttttc tttttgacac tgactggatg gggcgtgaag tttaccatgg ggatccaaat
1200
ggagatctga gtgagaggaa agcttatgac aatggtgtcc gagaaggtgc atttggtttg
1260
ctattgaatt cagttgtcct tggcgttggt tccttccttg ttgatccact atgccggatg
1320
attggtgcaa gattggtttg ggccattagc aacttcactg tgtttatttg catgatggct
1380
acaacaatac taagttggat ctcttctgat ctgtactcaa gcaaactcca tcacatcatt
1440
ggggcaaata aaacagtcaa gactacagca ttggttgttt tctctcttct cggactgcca
1500
ctctcgatca cttatagcgt tccattttct gtgactgctg agctgactgc tggtacagga
1560
ggtggacaag gtctggccac aggagtccta aatctcgcta ttgttgttcc ccagatagta
1620
gtgtcactcg gagcaggtcc atgggatgct ctatatgggg gaggaaacat cccagcgttc
1680
gccttggctt cggtcttctc cctggcggca ggtgtgctcg cagttctcaa gctaccaaag
1740
ctgtcgaact cgtaccaatc tgccggtttc catggatttg gctga
1785
<210> 288
<211> 594
<212> БЕЛОК
<213> Sorghum bicolor
<400> 288
Met Asp Ala Gly Thr Gly Gly Gly Gly Pro Thr Ala Ile Arg Val Pro
1 5 10 15
Tyr Arg His Leu Arg Asp Ala Glu Met Glu Leu Val Ser Leu Asn Gly
20 25 30
Ala Asp Ala Gly Pro Thr Pro His Lys Asp Ala Asp Gln Pro Arg Ser
35 40 45
Arg Gly Ala Asn Ala Asp Arg Thr Lys Leu Val Leu Ala Cys Met Val
50 55 60
Ala Ala Gly Val Gln Phe Gly Trp Ala Leu Gln Leu Ser Leu Leu Thr
65 70 75 80
Pro Tyr Ile Gln Thr Leu Gly Ile Asp His Ala Met Ala Ser Phe Ile
85 90 95
Trp Leu Cys Gly Pro Ile Thr Gly Phe Val Val Gln Pro Cys Val Gly
100 105 110
Val Trp Ser Asp Lys Cys Arg Ser Lys Tyr Gly Arg Arg Arg Pro Phe
115 120 125
Ile Leu Ala Gly Cys Ile Met Ile Cys Ala Ala Val Thr Leu Ile Gly
130 135 140
Phe Ser Ala Asp Leu Gly Tyr Ile Leu Gly Asp Thr Thr Glu His Cys
145 150 155 160
Arg Thr Tyr Lys Gly Ser Arg Phe Arg Ala Ala Met Val Phe Ile Leu
165 170 175
Gly Phe Trp Met Leu Asp Leu Ala Asn Asn Thr Val Gln Gly Pro Ala
180 185 190
Arg Ala Leu Leu Ala Asp Leu Ser Gly Pro Asp Gln Cys Asn Ser Ala
195 200 205
Asn Ala Ile Phe Cys Ser Trp Met Ala Val Gly Asn Ile Leu Gly Phe
210 215 220
Ser Ala Gly Ala Ser Gly Asp Trp His Lys Trp Phe Pro Phe Leu Met
225 230 235 240
Thr Arg Ala Cys Cys Glu Ala Cys Gly Asn Leu Lys Ala Ala Phe Leu
245 250 255
Val Ala Val Val Phe Leu Leu Phe Cys Met Ser Val Thr Leu Tyr Phe
260 265 270
Ala Glu Glu Ile Pro Leu Glu Pro Lys Asp Ala Gln Gly Leu Ser Asp
275 280 285
Ser Ala Pro Leu Leu Asn Gly Ser Arg Glu Asp Ala His Ala Leu Asn
290 295 300
Glu Pro Asn Asn Glu Arg Phe Pro Asn Gly His Val Asp Gly Asn Asn
305 310 315 320
Val Ser Ala Asn Asn Asn Thr Glu Glu Phe Pro Asn Ala Asn Ser Asn
325 330 335
Thr Asp Asn Gly Gly Val Phe Asn Asp Gly Pro Gly Ala Val Leu Val
340 345 350
Asn Ile Leu Thr Ser Met Arg His Leu Pro Pro Gly Met His Ser Val
355 360 365
Leu Val Val Met Ala Leu Thr Trp Leu Ser Trp Phe Pro Phe Phe Leu
370 375 380
Phe Asp Thr Asp Trp Met Gly Arg Glu Val Tyr His Gly Asp Pro Asn
385 390 395 400
Gly Asp Leu Ser Glu Arg Lys Ala Tyr Asp Asn Gly Val Arg Glu Gly
405 410 415
Ala Phe Gly Leu Leu Leu Asn Ser Val Val Leu Gly Val Gly Ser Phe
420 425 430
Leu Val Asp Pro Leu Cys Arg Met Ile Gly Ala Arg Leu Val Trp Ala
435 440 445
Ile Ser Asn Phe Thr Val Phe Ile Cys Met Met Ala Thr Thr Ile Leu
450 455 460
Ser Trp Ile Ser Ser Asp Leu Tyr Ser Ser Lys Leu His His Ile Ile
465 470 475 480
Gly Ala Asn Lys Thr Val Lys Thr Thr Ala Leu Val Val Phe Ser Leu
485 490 495
Leu Gly Leu Pro Leu Ser Ile Thr Tyr Ser Val Pro Phe Ser Val Thr
500 505 510
Ala Glu Leu Thr Ala Gly Thr Gly Gly Gly Gln Gly Leu Ala Thr Gly
515 520 525
Val Leu Asn Leu Ala Ile Val Val Pro Gln Ile Val Val Ser Leu Gly
530 535 540
Ala Gly Pro Trp Asp Ala Leu Tyr Gly Gly Gly Asn Ile Pro Ala Phe
545 550 555 560
Ala Leu Ala Ser Val Phe Ser Leu Ala Ala Gly Val Leu Ala Val Leu
565 570 575
Lys Leu Pro Lys Leu Ser Asn Ser Tyr Gln Ser Ala Gly Phe His Gly
580 585 590
Phe Gly
<210> 289
<211> 1779
<212> ДНК
<213> Zea mays
<400> 289
atggacgccg gcgccggggc cacggccatc cgagtgccct accgccacct ccgcgacgcc
60
gagatggagc tcgtcagcct caacggcggc gctcccggag gggacacggg cccgcccccg
120
ccaccgccca aggaccagcc gcggagccgc gccgacagga ccaagctcgt gctcgcctgc
180
atggtcgccg cgggcgtcca gttcggatgg gcgctgcagc tctccctcct tacgccatac
240
atccagaccc taggaataga ccatgccatg gcatcattta tttggttgtg tgggcctata
300
actggttttg tggttcaacc ttgtgttggt gtttggagtg ataagtgccg ttcaaagtat
360
gggaggagaa gaccatttat tttggctgga tgcataatga tatgtgctgc tgtaactcta
420
atcgggtttt ctgcagacct cggttacatc ttaggggaca ccactgagca ctgcagaaca
480
tataaaggtt caaggtttcg agctgctatt gttttcattc taggattctg gatgttggac
540
ctggcaaaca atacagtgca aggtcctgcg cgtgctcttc tagctgatct ttcaggtcct
600
gatcagtgca attctgcaaa tgcaatattc tgctcatgga tggctgttgg gaatattctt
660
ggtttttcag ctggagcgag tggggaatgg cacaagtggt ttccatttct aacgacaaga
720
gcatgctgtg aagcttgcgg taatttgaaa gcagctttct tagttgcagt tgtatttctt
780
ttgttatgta tgtctgttac cctgtacttc gctgaagaga gcccactaga tccaaaagat
840
acacaaggac tatcagattc tgctcctctg ctgaacggtt ctagagatgc tgcccatgca
900
tcaaatgaac caaataatga aagatttcct aatggccatg tgggtttaaa caatgtgtcg
960
gctaacaaca acactgagga atttacaaat gtgaattcca acacagagaa aggaggagtc
1020
ttcaatgatg ggccaggagc agttttggtt aacattttga ccagaatgag gcatctacct
1080
cctgggatgc attcagtgct tctagttatg gccctaacat ggttgtcatg gtttcccttt
1140
ttcctttttg acactgactg gatgggacgc gaagtttacc atggggatcc aaatggagat
1200
ttgagtgaga ggaaagctta tgacaatggt gtccgagaag gtgcatttgg tttgctattg
1260
aattcagttg tccttggcgt tggttcattc cttgttgatc cactatgccg gatgattggt
1320
gcaagattag tttgggccat tagcaacttc acggtgttta tttgcatgat ggctacaacg
1380
atattaagtt ggatctcttc tgatctgtac tcaagcaaac ttcatcacat catcggggca
1440
aataaaacag tcaagatcac ggcattggtt gttttctctc ttctcggatt gccactctcc
1500
atcacttaca gcgttccgtt ttctgtgact gctgagctga ctgccggtac aggaggtgga
1560
caaggtttgg ccacaggagt cctaaatctt gctatcgtgg ttccccagat agtagtgtcg
1620
cttggagcag gtccatggga cgctctgtat ggaggaggga ataccccggc gttcgtcttg
1680
gcttcggtct tctccctggc agcaggtgtg ctcgcagttc tcaagctgcc aaagctgtcc
1740
aactcgtacc aatctgccgg gttccatgga tttggctga
1779
<210> 290
<211> 592
<212> БЕЛОК
<213> Zea mays
<400> 290
Met Asp Ala Gly Ala Gly Ala Thr Ala Ile Arg Val Pro Tyr Arg His
1 5 10 15
Leu Arg Asp Ala Glu Met Glu Leu Val Ser Leu Asn Gly Gly Ala Pro
20 25 30
Gly Gly Asp Thr Gly Pro Pro Pro Pro Pro Pro Lys Asp Gln Pro Arg
35 40 45
Ser Arg Ala Asp Arg Thr Lys Leu Val Leu Ala Cys Met Val Ala Ala
50 55 60
Gly Val Gln Phe Gly Trp Ala Leu Gln Leu Ser Leu Leu Thr Pro Tyr
65 70 75 80
Ile Gln Thr Leu Gly Ile Asp His Ala Met Ala Ser Phe Ile Trp Leu
85 90 95
Cys Gly Pro Ile Thr Gly Phe Val Val Gln Pro Cys Val Gly Val Trp
100 105 110
Ser Asp Lys Cys Arg Ser Lys Tyr Gly Arg Arg Arg Pro Phe Ile Leu
115 120 125
Ala Gly Cys Ile Met Ile Cys Ala Ala Val Thr Leu Ile Gly Phe Ser
130 135 140
Ala Asp Leu Gly Tyr Ile Leu Gly Asp Thr Thr Glu His Cys Arg Thr
145 150 155 160
Tyr Lys Gly Ser Arg Phe Arg Ala Ala Ile Val Phe Ile Leu Gly Phe
165 170 175
Trp Met Leu Asp Leu Ala Asn Asn Thr Val Gln Gly Pro Ala Arg Ala
180 185 190
Leu Leu Ala Asp Leu Ser Gly Pro Asp Gln Cys Asn Ser Ala Asn Ala
195 200 205
Ile Phe Cys Ser Trp Met Ala Val Gly Asn Ile Leu Gly Phe Ser Ala
210 215 220
Gly Ala Ser Gly Glu Trp His Lys Trp Phe Pro Phe Leu Thr Thr Arg
225 230 235 240
Ala Cys Cys Glu Ala Cys Gly Asn Leu Lys Ala Ala Phe Leu Val Ala
245 250 255
Val Val Phe Leu Leu Leu Cys Met Ser Val Thr Leu Tyr Phe Ala Glu
260 265 270
Glu Ser Pro Leu Asp Pro Lys Asp Thr Gln Gly Leu Ser Asp Ser Ala
275 280 285
Pro Leu Leu Asn Gly Ser Arg Asp Ala Ala His Ala Ser Asn Glu Pro
290 295 300
Asn Asn Glu Arg Phe Pro Asn Gly His Val Gly Leu Asn Asn Val Ser
305 310 315 320
Ala Asn Asn Asn Thr Glu Glu Phe Thr Asn Val Asn Ser Asn Thr Glu
325 330 335
Lys Gly Gly Val Phe Asn Asp Gly Pro Gly Ala Val Leu Val Asn Ile
340 345 350
Leu Thr Arg Met Arg His Leu Pro Pro Gly Met His Ser Val Leu Leu
355 360 365
Val Met Ala Leu Thr Trp Leu Ser Trp Phe Pro Phe Phe Leu Phe Asp
370 375 380
Thr Asp Trp Met Gly Arg Glu Val Tyr His Gly Asp Pro Asn Gly Asp
385 390 395 400
Leu Ser Glu Arg Lys Ala Tyr Asp Asn Gly Val Arg Glu Gly Ala Phe
405 410 415
Gly Leu Leu Leu Asn Ser Val Val Leu Gly Val Gly Ser Phe Leu Val
420 425 430
Asp Pro Leu Cys Arg Met Ile Gly Ala Arg Leu Val Trp Ala Ile Ser
435 440 445
Asn Phe Thr Val Phe Ile Cys Met Met Ala Thr Thr Ile Leu Ser Trp
450 455 460
Ile Ser Ser Asp Leu Tyr Ser Ser Lys Leu His His Ile Ile Gly Ala
465 470 475 480
Asn Lys Thr Val Lys Ile Thr Ala Leu Val Val Phe Ser Leu Leu Gly
485 490 495
Leu Pro Leu Ser Ile Thr Tyr Ser Val Pro Phe Ser Val Thr Ala Glu
500 505 510
Leu Thr Ala Gly Thr Gly Gly Gly Gln Gly Leu Ala Thr Gly Val Leu
515 520 525
Asn Leu Ala Ile Val Val Pro Gln Ile Val Val Ser Leu Gly Ala Gly
530 535 540
Pro Trp Asp Ala Leu Tyr Gly Gly Gly Asn Thr Pro Ala Phe Val Leu
545 550 555 560
Ala Ser Val Phe Ser Leu Ala Ala Gly Val Leu Ala Val Leu Lys Leu
565 570 575
Pro Lys Leu Ser Asn Ser Tyr Gln Ser Ala Gly Phe His Gly Phe Gly
580 585 590
<210> 291
<211> 693
<212> ДНК
<213> Arabidopsis thaliana
<400> 291
atggcagact tgagttttta tgtcggagtc atagggaatg ttatatcggt gcttgtcttc
60
ctctcccctg tggagacgtt ttggaggata gtgcagcgga gatcgacgga ggaatacgag
120
tgttttccgt acatttgcac gttaatgagc tcgtcgttat ggacatatta cggaatagtg
180
acacctggtg aatacttggt ttctactgtc aatggctttg gtgctcttgc tgaatccatc
240
tacgttctca ttttcctctt ctttgtcccc aaatccagat tcttgaaaac agttgttgtg
300
gttctagctt tgaacgtgtg tttcccagtt atcgcgattg cgggaacaag aactctgttt
360
ggagatgcaa actcgcgttc tagttcaatg ggtttcatat gtgctactct caacattatc
420
atgtatggtt ctcctctttc agctattaaa acggttgtga cgacgagaag tgtgcagttt
480
atgccatttt ggttgtcatt tttcctcttt ctgaacggcg cgatttgggg tgtctacgct
540
ttactcctac acgatatgtt tctactagtg ccaaatggaa tgggattctt tcttgggata
600
atgcaactcc taatttatgc ctattacaga aatgccgaac caattgtaga agatgaagaa
660
ggcctcatac caaatcaacc tctcctcgct taa
693
<210> 292
<211> 230
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 292
Met Ala Asp Leu Ser Phe Tyr Val Gly Val Ile Gly Asn Val Ile Ser
1 5 10 15
Val Leu Val Phe Leu Ser Pro Val Glu Thr Phe Trp Arg Ile Val Gln
20 25 30
Arg Arg Ser Thr Glu Glu Tyr Glu Cys Phe Pro Tyr Ile Cys Thr Leu
35 40 45
Met Ser Ser Ser Leu Trp Thr Tyr Tyr Gly Ile Val Thr Pro Gly Glu
50 55 60
Tyr Leu Val Ser Thr Val Asn Gly Phe Gly Ala Leu Ala Glu Ser Ile
65 70 75 80
Tyr Val Leu Ile Phe Leu Phe Phe Val Pro Lys Ser Arg Phe Leu Lys
85 90 95
Thr Val Val Val Val Leu Ala Leu Asn Val Cys Phe Pro Val Ile Ala
100 105 110
Ile Ala Gly Thr Arg Thr Leu Phe Gly Asp Ala Asn Ser Arg Ser Ser
115 120 125
Ser Met Gly Phe Ile Cys Ala Thr Leu Asn Ile Ile Met Tyr Gly Ser
130 135 140
Pro Leu Ser Ala Ile Lys Thr Val Val Thr Thr Arg Ser Val Gln Phe
145 150 155 160
Met Pro Phe Trp Leu Ser Phe Phe Leu Phe Leu Asn Gly Ala Ile Trp
165 170 175
Gly Val Tyr Ala Leu Leu Leu His Asp Met Phe Leu Leu Val Pro Asn
180 185 190
Gly Met Gly Phe Phe Leu Gly Ile Met Gln Leu Leu Ile Tyr Ala Tyr
195 200 205
Tyr Arg Asn Ala Glu Pro Ile Val Glu Asp Glu Glu Gly Leu Ile Pro
210 215 220
Asn Gln Pro Leu Leu Ala
225 230
<210> 293
<211> 4008
<212> ДНК
<213> Arabidopsis thaliana
<400> 293
atggataata acaattggag gccttctctt ccaaacggag agcctgccat ggatacaggt
60
gactggagaa cgcaattgcc acctgattct cgtcagaaga tcgtcaacaa gataatggaa
120
acactaaaga agcaccttcc attttccgga ccagagggaa ttaacgagct caggagaatt
180
gcagctagat tcgaggagaa aattttcagc ggtgctctta accagactga ttaccttcgg
240
aaaatatcaa tgaagatgct gacaatggag actaaatcac aaaatgcagc tggttcttcc
300
gcagctatcc ctgccgctaa taatggcaca tccatagatt cgatacccac caatcaaggc
360
caacttcttc caggatcgtt atcaaccaat caatctcaag cacctcagcc gttgctgtcc
420
caaaccatgc agaataatac tgcctctgga atgacgggtt ctactgcttt accatcttcc
480
atgccacctg tttcttccat aaccaataat aacaccacaa gcgttgtgaa ccagaatgcc
540
aatatgcaaa atgtagctgg aatgttgcaa gattcatctg ggcagcatgg cctttcctcg
600
aacatgtttt caggacccca aaggcagatg ctgggtaggc cacatgctat gtcttcacag
660
caacaacagc agccatatct ttaccagcag cagctacagc agcaacttct caagcagaac
720
ttccagtcag ggaatgttcc caatcccaat tcgcttttgc catcacacat tcaacaacag
780
cagcaaaatg tgctgcagcc taatcaactg cattcatctc aacaacctgg tgttccaaca
840
tctgcgactc agccctccac tgtgaactca gctcctctcc aaggtctcca caccaatcag
900
caatcaagtc cgcaattgtc ttctcagcag acgacacaat ctatgcttcg tcagcatcaa
960
tcgtcgatgc taaggcaaca tccgcaatca caacaagcct ctggtatcca tcagcagcaa
1020
tcttcattgc cgcaacaatc aatttctcct ctacagcagc agcctacaca attaatgcgg
1080
caacaagctg caaatagctc aggcatccaa cagaagcaga tgatggggca gcatgttgtt
1140
ggggatatgc agcagcaaca tcagcaaagg ttactgaacc aacaaaataa tgttatgaac
1200
atacaacagc agcagtcgca gcagcaacca ctgcagcagc cacagcaaca gcagaaacag
1260
cagccaccgg cccagcagca gttgatgtct caacaaaaca gcctccaggc aacgcatcag
1320
aacccactgg gcactcaaag caatgttgca ggattgcagc aaccacaaca acagatgctc
1380
aattcccagg ttggcaattc gagtttgcag aataaccagc actcggtgca catgttatca
1440
caaccaacgg ttgggctgca acgaacacat caggctggcc atggcttgta ttcttctcag
1500
ggacaacagt cacaaaatca gccatcacaa cagcagatga tgccacagct tcaatcgcat
1560
catcagcagc tgggtttgca acaacaacct aatctgctac aacaggatgt gcaacaaagg
1620
ctacaagctt caggccaagt cactggttcc ctgcttccac ctcaaaatgt tgtggaccaa
1680
cagagacaac tatatcaatc ccaaagaacc cttccggaga tgccatcatc atcgctggat
1740
tcgacagcac agacggaaag tgcaaatgga ggtgattggc aagaggaggt ttaccaaaag
1800
atcaaatcta tgaaagagac ttacttacca gatctgaatg aaatctacca gagagttgca
1860
gcaaagttgc agcaagattc tatgccacag caacaaagat cagatcagct tgagaaactg
1920
agacaattca aaacaatgtt ggagcgaatg atacaatttc tatctgtttc aaagagcaac
1980
atcatgcccg cattaaaaga taaggtggct tattatgaga agcagattat aggtttctta
2040
aatatgcaca ggccgaggaa gccagtacag caagggcagc ttccgcaatc tcagatgcag
2100
cctatgcagc aaccacaatc tcagacagtt caagatcaat cccatgataa ccaaacaaat
2160
ccgcagatgc aatcaatgag catgcagggt gctgggccaa gggcacaaca gagtagtatg
2220
acaaatatgc agagcaatgt tctatcatct cgtcctggag tttcagctcc acagcagaac
2280
attcccagtt ccataccggc ttctagttta gaatcaggcc aaggaaatac cttgaacaat
2340
ggacagcagg ttgccatggg atctatgcaa caaaatactt ctcaactagt aaataacagt
2400
tctgcctctg ctcaaagtgg gttgagcaca ctgcagtcga atgtgaatca accccagtta
2460
agttccagtt tgcttcagca tcagcacctc aagcaacagc aagatcaaca aatgcagctc
2520
aaacagcaat ttcaacagcg ccagatgcaa cagcaacagt tgcaagcaag acagcaacag
2580
caacagcaac agttgcaagc aagacagcaa gcggcacaat tacagcagat gaatgatatg
2640
aatgatttaa catcgaggca ggggatgaat gtcagtcgtg ggatgtttca gcaacattct
2700
atgcagggtc agcgtgcgaa ttatcctctt caacagttaa aaccaggagc tgtttcgtcg
2760
cctcaacttc tgcaaggtgc atctcctcag atgtcacaac atttgtctcc tcaggttgac
2820
cagaaaaata ctgtcaacaa gatgggaact ccattgcaac ctgcaaattc cccttttgtt
2880
gtcccatctc cttcttcaac ccccttggct ccgtccccta tgcaagttga ctctgagaaa
2940
cctggttctt cttcgttgtc aatgggaaat attgcacgcc aacaagcaac cggcatgcaa
3000
ggtgtagttc agtccctagc aattggcact ccagggatct ctgcctctcc tctccttcag
3060
gagtttacta gtcctgatgg gaatatttta aattcttcga caattacatc tggaaaaccg
3120
agtgctactg agctgcctat tgaacgcctt attagagccg tgaagtccat ctcaccacaa
3180
gccctttctt ctgcagtaag tgacatcgga tctgttgtaa gcatggttga tcggatagct
3240
ggttcagcac caggaaacgg ttcaagagct tccgttggtg aagacttggt tgcaatgact
3300
aagtgccgtc tccaagcaag aaacttcatg acccaagagg gaatgatggc gactaagaaa
3360
atgaagcgtc acacaaccgc aatgccacta agcgttgctt cactgggagg aagtgttggt
3420
gataactaca agcagtttgc tggttcagaa acatccgatc tagaatctac tgcgacttcg
3480
gatggcaaga aggcaagaac tgagaccgag catgcccttt tggaggaaat aaaggaaata
3540
aaccagcggc tgatagatac agttgttgag ataagtgatg atgaagatgc tgctgatcct
3600
agtgaggttg caatatcaag catagggtgt gaaggaacaa cagttagatt ctcgtttata
3660
gctgtttctc tcagcccagc cttgaaggct catctctcat caacacaaat gtctcctatt
3720
caaccattgc gtctgctggt tccttgtagc taccccaatg gctctccatc tcttctagat
3780
aaactcccgg tcgaaaccag caaagaaaac gaggacctgt cgtccaaagc tatggcaagg
3840
ttcaacatat tgctaagaag tttgtcacag ccgatgtcgc tcaaagacat agccaagaca
3900
tgggacgcct gtgctcgggc agtgatctgt gagtacgcac agcaatttgg cggtggaact
3960
ttcagctcaa aatacggcac ttgggagaaa tatgtagcag cttcctga
4008
<210> 294
<211> 1335
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 294
Met Asp Asn Asn Asn Trp Arg Pro Ser Leu Pro Asn Gly Glu Pro Ala
1 5 10 15
Met Asp Thr Gly Asp Trp Arg Thr Gln Leu Pro Pro Asp Ser Arg Gln
20 25 30
Lys Ile Val Asn Lys Ile Met Glu Thr Leu Lys Lys His Leu Pro Phe
35 40 45
Ser Gly Pro Glu Gly Ile Asn Glu Leu Arg Arg Ile Ala Ala Arg Phe
50 55 60
Glu Glu Lys Ile Phe Ser Gly Ala Leu Asn Gln Thr Asp Tyr Leu Arg
65 70 75 80
Lys Ile Ser Met Lys Met Leu Thr Met Glu Thr Lys Ser Gln Asn Ala
85 90 95
Ala Gly Ser Ser Ala Ala Ile Pro Ala Ala Asn Asn Gly Thr Ser Ile
100 105 110
Asp Ser Ile Pro Thr Asn Gln Gly Gln Leu Leu Pro Gly Ser Leu Ser
115 120 125
Thr Asn Gln Ser Gln Ala Pro Gln Pro Leu Leu Ser Gln Thr Met Gln
130 135 140
Asn Asn Thr Ala Ser Gly Met Thr Gly Ser Thr Ala Leu Pro Ser Ser
145 150 155 160
Met Pro Pro Val Ser Ser Ile Thr Asn Asn Asn Thr Thr Ser Val Val
165 170 175
Asn Gln Asn Ala Asn Met Gln Asn Val Ala Gly Met Leu Gln Asp Ser
180 185 190
Ser Gly Gln His Gly Leu Ser Ser Asn Met Phe Ser Gly Pro Gln Arg
195 200 205
Gln Met Leu Gly Arg Pro His Ala Met Ser Ser Gln Gln Gln Gln Gln
210 215 220
Pro Tyr Leu Tyr Gln Gln Gln Leu Gln Gln Gln Leu Leu Lys Gln Asn
225 230 235 240
Phe Gln Ser Gly Asn Val Pro Asn Pro Asn Ser Leu Leu Pro Ser His
245 250 255
Ile Gln Gln Gln Gln Gln Asn Val Leu Gln Pro Asn Gln Leu His Ser
260 265 270
Ser Gln Gln Pro Gly Val Pro Thr Ser Ala Thr Gln Pro Ser Thr Val
275 280 285
Asn Ser Ala Pro Leu Gln Gly Leu His Thr Asn Gln Gln Ser Ser Pro
290 295 300
Gln Leu Ser Ser Gln Gln Thr Thr Gln Ser Met Leu Arg Gln His Gln
305 310 315 320
Ser Ser Met Leu Arg Gln His Pro Gln Ser Gln Gln Ala Ser Gly Ile
325 330 335
His Gln Gln Gln Ser Ser Leu Pro Gln Gln Ser Ile Ser Pro Leu Gln
340 345 350
Gln Gln Pro Thr Gln Leu Met Arg Gln Gln Ala Ala Asn Ser Ser Gly
355 360 365
Ile Gln Gln Lys Gln Met Met Gly Gln His Val Val Gly Asp Met Gln
370 375 380
Gln Gln His Gln Gln Arg Leu Leu Asn Gln Gln Asn Asn Val Met Asn
385 390 395 400
Ile Gln Gln Gln Gln Ser Gln Gln Gln Pro Leu Gln Gln Pro Gln Gln
405 410 415
Gln Gln Lys Gln Gln Pro Pro Ala Gln Gln Gln Leu Met Ser Gln Gln
420 425 430
Asn Ser Leu Gln Ala Thr His Gln Asn Pro Leu Gly Thr Gln Ser Asn
435 440 445
Val Ala Gly Leu Gln Gln Pro Gln Gln Gln Met Leu Asn Ser Gln Val
450 455 460
Gly Asn Ser Ser Leu Gln Asn Asn Gln His Ser Val His Met Leu Ser
465 470 475 480
Gln Pro Thr Val Gly Leu Gln Arg Thr His Gln Ala Gly His Gly Leu
485 490 495
Tyr Ser Ser Gln Gly Gln Gln Ser Gln Asn Gln Pro Ser Gln Gln Gln
500 505 510
Met Met Pro Gln Leu Gln Ser His His Gln Gln Leu Gly Leu Gln Gln
515 520 525
Gln Pro Asn Leu Leu Gln Gln Asp Val Gln Gln Arg Leu Gln Ala Ser
530 535 540
Gly Gln Val Thr Gly Ser Leu Leu Pro Pro Gln Asn Val Val Asp Gln
545 550 555 560
Gln Arg Gln Leu Tyr Gln Ser Gln Arg Thr Leu Pro Glu Met Pro Ser
565 570 575
Ser Ser Leu Asp Ser Thr Ala Gln Thr Glu Ser Ala Asn Gly Gly Asp
580 585 590
Trp Gln Glu Glu Val Tyr Gln Lys Ile Lys Ser Met Lys Glu Thr Tyr
595 600 605
Leu Pro Asp Leu Asn Glu Ile Tyr Gln Arg Val Ala Ala Lys Leu Gln
610 615 620
Gln Asp Ser Met Pro Gln Gln Gln Arg Ser Asp Gln Leu Glu Lys Leu
625 630 635 640
Arg Gln Phe Lys Thr Met Leu Glu Arg Met Ile Gln Phe Leu Ser Val
645 650 655
Ser Lys Ser Asn Ile Met Pro Ala Leu Lys Asp Lys Val Ala Tyr Tyr
660 665 670
Glu Lys Gln Ile Ile Gly Phe Leu Asn Met His Arg Pro Arg Lys Pro
675 680 685
Val Gln Gln Gly Gln Leu Pro Gln Ser Gln Met Gln Pro Met Gln Gln
690 695 700
Pro Gln Ser Gln Thr Val Gln Asp Gln Ser His Asp Asn Gln Thr Asn
705 710 715 720
Pro Gln Met Gln Ser Met Ser Met Gln Gly Ala Gly Pro Arg Ala Gln
725 730 735
Gln Ser Ser Met Thr Asn Met Gln Ser Asn Val Leu Ser Ser Arg Pro
740 745 750
Gly Val Ser Ala Pro Gln Gln Asn Ile Pro Ser Ser Ile Pro Ala Ser
755 760 765
Ser Leu Glu Ser Gly Gln Gly Asn Thr Leu Asn Asn Gly Gln Gln Val
770 775 780
Ala Met Gly Ser Met Gln Gln Asn Thr Ser Gln Leu Val Asn Asn Ser
785 790 795 800
Ser Ala Ser Ala Gln Ser Gly Leu Ser Thr Leu Gln Ser Asn Val Asn
805 810 815
Gln Pro Gln Leu Ser Ser Ser Leu Leu Gln His Gln His Leu Lys Gln
820 825 830
Gln Gln Asp Gln Gln Met Gln Leu Lys Gln Gln Phe Gln Gln Arg Gln
835 840 845
Met Gln Gln Gln Gln Leu Gln Ala Arg Gln Gln Gln Gln Gln Gln Gln
850 855 860
Leu Gln Ala Arg Gln Gln Ala Ala Gln Leu Gln Gln Met Asn Asp Met
865 870 875 880
Asn Asp Leu Thr Ser Arg Gln Gly Met Asn Val Ser Arg Gly Met Phe
885 890 895
Gln Gln His Ser Met Gln Gly Gln Arg Ala Asn Tyr Pro Leu Gln Gln
900 905 910
Leu Lys Pro Gly Ala Val Ser Ser Pro Gln Leu Leu Gln Gly Ala Ser
915 920 925
Pro Gln Met Ser Gln His Leu Ser Pro Gln Val Asp Gln Lys Asn Thr
930 935 940
Val Asn Lys Met Gly Thr Pro Leu Gln Pro Ala Asn Ser Pro Phe Val
945 950 955 960
Val Pro Ser Pro Ser Ser Thr Pro Leu Ala Pro Ser Pro Met Gln Val
965 970 975
Asp Ser Glu Lys Pro Gly Ser Ser Ser Leu Ser Met Gly Asn Ile Ala
980 985 990
Arg Gln Gln Ala Thr Gly Met Gln Gly Val Val Gln Ser Leu Ala Ile
995 1000 1005
Gly Thr Pro Gly Ile Ser Ala Ser Pro Leu Leu Gln Glu Phe Thr
1010 1015 1020
Ser Pro Asp Gly Asn Ile Leu Asn Ser Ser Thr Ile Thr Ser Gly
1025 1030 1035
Lys Pro Ser Ala Thr Glu Leu Pro Ile Glu Arg Leu Ile Arg Ala
1040 1045 1050
Val Lys Ser Ile Ser Pro Gln Ala Leu Ser Ser Ala Val Ser Asp
1055 1060 1065
Ile Gly Ser Val Val Ser Met Val Asp Arg Ile Ala Gly Ser Ala
1070 1075 1080
Pro Gly Asn Gly Ser Arg Ala Ser Val Gly Glu Asp Leu Val Ala
1085 1090 1095
Met Thr Lys Cys Arg Leu Gln Ala Arg Asn Phe Met Thr Gln Glu
1100 1105 1110
Gly Met Met Ala Thr Lys Lys Met Lys Arg His Thr Thr Ala Met
1115 1120 1125
Pro Leu Ser Val Ala Ser Leu Gly Gly Ser Val Gly Asp Asn Tyr
1130 1135 1140
Lys Gln Phe Ala Gly Ser Glu Thr Ser Asp Leu Glu Ser Thr Ala
1145 1150 1155
Thr Ser Asp Gly Lys Lys Ala Arg Thr Glu Thr Glu His Ala Leu
1160 1165 1170
Leu Glu Glu Ile Lys Glu Ile Asn Gln Arg Leu Ile Asp Thr Val
1175 1180 1185
Val Glu Ile Ser Asp Asp Glu Asp Ala Ala Asp Pro Ser Glu Val
1190 1195 1200
Ala Ile Ser Ser Ile Gly Cys Glu Gly Thr Thr Val Arg Phe Ser
1205 1210 1215
Phe Ile Ala Val Ser Leu Ser Pro Ala Leu Lys Ala His Leu Ser
1220 1225 1230
Ser Thr Gln Met Ser Pro Ile Gln Pro Leu Arg Leu Leu Val Pro
1235 1240 1245
Cys Ser Tyr Pro Asn Gly Ser Pro Ser Leu Leu Asp Lys Leu Pro
1250 1255 1260
Val Glu Thr Ser Lys Glu Asn Glu Asp Leu Ser Ser Lys Ala Met
1265 1270 1275
Ala Arg Phe Asn Ile Leu Leu Arg Ser Leu Ser Gln Pro Met Ser
1280 1285 1290
Leu Lys Asp Ile Ala Lys Thr Trp Asp Ala Cys Ala Arg Ala Val
1295 1300 1305
Ile Cys Glu Tyr Ala Gln Gln Phe Gly Gly Gly Thr Phe Ser Ser
1310 1315 1320
Lys Tyr Gly Thr Trp Glu Lys Tyr Val Ala Ala Ser
1325 1330 1335
<210> 295
<211> 3927
<212> ДНК
<213> Zea mays
<400> 295
atggacagcg ccgccaactg gcggtcaacg cagggcaccg accctgccgc cggcggcgtc
60
gatccaaacg cggctgcccc cgccggaagc gactggcgca cccagcttca gccagaggcg
120
cgccacagga tcgtgaataa gataatggag actctgaaga agcacctgcc agtatcagta
180
ccagaggggc tgactgagct tcacaaaatt gctgtgcgtt ttgaagagaa aatctatact
240
gcagccacca gccagtctga ttatttgcgg aagatttcgc tgaaaatgct gtccatggaa
300
agtcagacaa agacacaaca gaaccctgga aatgttcaag tgattccaaa tcaaaaccct
360
cctggtccag cacctggcct tcctccacaa gttagtaatc cagcacagtc atcagctatc
420
ccattgattt ctcagcaaca gacacggcaa tcaaatgcct ctacatctgt tcaaggttct
480
cttcctagtc ttggtcagaa ctcgtcgagt gtcagccagg catcgacgct gcataatatg
540
tctgtcatgc cacaaaatac catgaacaat ggtttagcac aaggtactcc acaagatatg
600
tatgctgcac aaaggcaaat ggctggtagg cagcagcaac aacaacaaca acaagcacat
660
aatcagttaa tttatcaaca acagaaatac ttgaaccaga aattgcagca gaattcactt
720
atgccatcgc acattcagca gcagcaacct ttattgcagt caacacagat gcaatcttca
780
cagcagccca tgatgcaaat gtcatctggt cttcagcctg ggcagactgc cattccacaa
840
actcagtcca tgacgatgca ttcagctaca caaagtggta ttcaacaaaa cccattgaat
900
tcagtacaac aatctgtgca accattactc catcagccta cacaatctgt agtgaggcag
960
cagcaacatc cacagtccat gcaccagcag tcttctctgc agcaaactca gccaactcaa
1020
cagcccaaca ttcctttgca acaacaacaa ccacaattaa tgaaccagca gtcaaattta
1080
cagcaaaatc agttaatgaa tcaacagagt ggtgttgtgg agacgcaaca gcaacaaagg
1140
ctgccagttc agtcaaataa tcttttgaat atgcagcaaa cacagcagat gatgaaccag
1200
caatctatgt ccttgcacca gccacaacaa ttggcaaacc aaggaaatat gtcaagtcta
1260
catcagcagc agcatcagca aaatcagcag cagcagcagc ttctcggaac tgggccaaat
1320
gcccgtatgc atatgttaca gcagcaaaag gtaattcagc aaccacagca gcaacagcat
1380
gctcagcaga catcaatggg tttgatacaa cctcagtctc agcagaacca acttcagcaa
1440
cctcagcaac atatgatgtc gcagttccag tctcagccta atcaaatgca gcaacaattg
1500
ggaatgcaac aaaggctcca aacttcagct ggcatgctct tacaacaaaa taacattgat
1560
caaaagcaat atgttcaggc acagaggggc cttcaagaag cacccagtac atctgtggat
1620
tccactgctc aaactggtca tccaggcata ggtgatttgc aagaggagtt atatcaaatg
1680
attaagagtt taaaggacca atactttgtg gaattgaatg aattgtacaa taaggtatct
1740
atcaagatac aacagattga caaccaaatg cctgctcaaa agtcagcaga gcagtatgaa
1800
aagatgaagg gctttaaagg aatgctggag cgcactttgc atttcctgca agttaacaag
1860
agcaacattc atccaggttt gagggaaaaa atccccattt acgagaggca aattctcagt
1920
atcctaagtt cacaaagaag gaaacctgtg caggcacctg ggcagcaaac gtttcagcaa
1980
tctagtgggc aagctcctag ctctaacatt tcacagcaac ttcagacttc ccaaggtttg
2040
cagcagcatg atagtcatac tagtcagatg cctcaagcaa gtttaccaag tatgaacaca
2100
ggagtacaaa cctctggagc acctgcgcct caaggaacaa actttggtgt tccaacaaca
2160
cagcaaaatg tcacaaatgc accacaggct ggctctaatt tggagaatgc tcagggaaat
2220
aattttaatc atgtgcagca tggttcaatg ggtgctgctt tgcaacagga aaggactggt
2280
cccatgcagg gtgcattgaa tgcacagcag cagtccagta gcaacatgat atccaacaat
2340
gcaatgagta caatgcagac taataacaat gcaaatgcga attcattgca gcagttaaag
2400
cagcaacgtc aggagcatca aatgatgcaa agtcagcaaa tgaagcagcg ccagcagatg
2460
atgcaacaga tacaacaaaa gcaaacgctt caaccgcagc tcccaataca gcaactaaag
2520
aaacaacagc agcaagggca gatgcagttt ccacagcttc attctggaaa tgatgtgaat
2580
gagctaaagg ttaggcaagg agctgcaatc aaatctggaa tgtatcaaca gctgagccag
2640
cgtaactatt atcagcagat aaaacagggt ggtgtctttc caatttcttc tccgcaaacc
2700
ctccaaacat cgtccccaca aatttcacac cattctcctc aggttgatca gcacagtctg
2760
ttgcaatctc aagtcaaaac cgggacacca ttgcattcag ctaactcacc atttgtccca
2820
tctccatccc ctcctgtggc cccatcaccg atgccaatgg attcggataa accactatcc
2880
aacttatctt cagttactag tgctgggcag gctggacatc agcaaacatc tcttgcacct
2940
caaacacaat ctatagccgt taacacacca ggtatatcag cgtcaccctt gcttgcagaa
3000
ttcacaagtg ctgatggaag tccggccaat gtaccaactc aagttccagc caaatcaagc
3060
gcagcagaaa ggcctctgga ccgtttgctt aaagcattgc gaacaacaca gcgtgagtcc
3120
ctaagtgctg cagtcagcga tattggatct gtcgtgagta tgattgacag gattgctgga
3180
tcagcgcctg gtaatggttc tagagctgct gtaggagaag atcttgtcgc tatgacaaag
3240
tgccgcttgc aagccaggaa cttcataact catgacggga gtggtgcatc aaagaaaatg
3300
aagcgggaca cgagtgctat gcctcttaat gtgtcatcag ctggaagcgt gaatgatagc
3360
ttgaagcaat catatggtgt tggtactcca gagctacaat caactgcaac ctcacgtgtc
3420
aagtggcaaa gggctgaggt aaaccatgct ctgatggagg agattcagga gataaaccaa
3480
cagcttatag atacagagct ccatgtctct gaggatgatg ctgagtcctt cactacatct
3540
gaggggggca aagggacggt cattagatgc acattcactg ctgttgctgt ttgccccagc
3600
ttgaaatccg tgtttgcgtc agcgcagatg agtccaattt tgccgttgag gttgcttgtt
3660
cctgctagct acccgaaatg ctccccggtg cttctagaca agtttcctga tgaacaatgc
3720
aggaactcag acgacctgtc taccaaggcc aagacaaagt tcagcgtatt gctccggggt
3780
ctagctgagc ccatgtcact gcgagaaatc gcaaggacat gggatgcttg cgctcgcaaa
3840
gtgatcacag agtatgccca gcaaaccgga ggaggcagtt tcagctcgag ctatggttgc
3900
tgggaaagct gcgtaggcgc ttgttaa
3927
<210> 296
<211> 1308
<212> БЕЛОК
<213> Zea mays
<400> 296
Met Asp Ser Ala Ala Asn Trp Arg Ser Thr Gln Gly Thr Asp Pro Ala
1 5 10 15
Ala Gly Gly Val Asp Pro Asn Ala Ala Ala Pro Ala Gly Ser Asp Trp
20 25 30
Arg Thr Gln Leu Gln Pro Glu Ala Arg His Arg Ile Val Asn Lys Ile
35 40 45
Met Glu Thr Leu Lys Lys His Leu Pro Val Ser Val Pro Glu Gly Leu
50 55 60
Thr Glu Leu His Lys Ile Ala Val Arg Phe Glu Glu Lys Ile Tyr Thr
65 70 75 80
Ala Ala Thr Ser Gln Ser Asp Tyr Leu Arg Lys Ile Ser Leu Lys Met
85 90 95
Leu Ser Met Glu Ser Gln Thr Lys Thr Gln Gln Asn Pro Gly Asn Val
100 105 110
Gln Val Ile Pro Asn Gln Asn Pro Pro Gly Pro Ala Pro Gly Leu Pro
115 120 125
Pro Gln Val Ser Asn Pro Ala Gln Ser Ser Ala Ile Pro Leu Ile Ser
130 135 140
Gln Gln Gln Thr Arg Gln Ser Asn Ala Ser Thr Ser Val Gln Gly Ser
145 150 155 160
Leu Pro Ser Leu Gly Gln Asn Ser Ser Ser Val Ser Gln Ala Ser Thr
165 170 175
Leu His Asn Met Ser Val Met Pro Gln Asn Thr Met Asn Asn Gly Leu
180 185 190
Ala Gln Gly Thr Pro Gln Asp Met Tyr Ala Ala Gln Arg Gln Met Ala
195 200 205
Gly Arg Gln Gln Gln Gln Gln Gln Gln Gln Ala His Asn Gln Leu Ile
210 215 220
Tyr Gln Gln Gln Lys Tyr Leu Asn Gln Lys Leu Gln Gln Asn Ser Leu
225 230 235 240
Met Pro Ser His Ile Gln Gln Gln Gln Pro Leu Leu Gln Ser Thr Gln
245 250 255
Met Gln Ser Ser Gln Gln Pro Met Met Gln Met Ser Ser Gly Leu Gln
260 265 270
Pro Gly Gln Thr Ala Ile Pro Gln Thr Gln Ser Met Thr Met His Ser
275 280 285
Ala Thr Gln Ser Gly Ile Gln Gln Asn Pro Leu Asn Ser Val Gln Gln
290 295 300
Ser Val Gln Pro Leu Leu His Gln Pro Thr Gln Ser Val Val Arg Gln
305 310 315 320
Gln Gln His Pro Gln Ser Met His Gln Gln Ser Ser Leu Gln Gln Thr
325 330 335
Gln Pro Thr Gln Gln Pro Asn Ile Pro Leu Gln Gln Gln Gln Pro Gln
340 345 350
Leu Met Asn Gln Gln Ser Asn Leu Gln Gln Asn Gln Leu Met Asn Gln
355 360 365
Gln Ser Gly Val Val Glu Thr Gln Gln Gln Gln Arg Leu Pro Val Gln
370 375 380
Ser Asn Asn Leu Leu Asn Met Gln Gln Thr Gln Gln Met Met Asn Gln
385 390 395 400
Gln Ser Met Ser Leu His Gln Pro Gln Gln Leu Ala Asn Gln Gly Asn
405 410 415
Met Ser Ser Leu His Gln Gln Gln His Gln Gln Asn Gln Gln Gln Gln
420 425 430
Gln Leu Leu Gly Thr Gly Pro Asn Ala Arg Met His Met Leu Gln Gln
435 440 445
Gln Lys Val Ile Gln Gln Pro Gln Gln Gln Gln His Ala Gln Gln Thr
450 455 460
Ser Met Gly Leu Ile Gln Pro Gln Ser Gln Gln Asn Gln Leu Gln Gln
465 470 475 480
Pro Gln Gln His Met Met Ser Gln Phe Gln Ser Gln Pro Asn Gln Met
485 490 495
Gln Gln Gln Leu Gly Met Gln Gln Arg Leu Gln Thr Ser Ala Gly Met
500 505 510
Leu Leu Gln Gln Asn Asn Ile Asp Gln Lys Gln Tyr Val Gln Ala Gln
515 520 525
Arg Gly Leu Gln Glu Ala Pro Ser Thr Ser Val Asp Ser Thr Ala Gln
530 535 540
Thr Gly His Pro Gly Ile Gly Asp Leu Gln Glu Glu Leu Tyr Gln Met
545 550 555 560
Ile Lys Ser Leu Lys Asp Gln Tyr Phe Val Glu Leu Asn Glu Leu Tyr
565 570 575
Asn Lys Val Ser Ile Lys Ile Gln Gln Ile Asp Asn Gln Met Pro Ala
580 585 590
Gln Lys Ser Ala Glu Gln Tyr Glu Lys Met Lys Gly Phe Lys Gly Met
595 600 605
Leu Glu Arg Thr Leu His Phe Leu Gln Val Asn Lys Ser Asn Ile His
610 615 620
Pro Gly Leu Arg Glu Lys Ile Pro Ile Tyr Glu Arg Gln Ile Leu Ser
625 630 635 640
Ile Leu Ser Ser Gln Arg Arg Lys Pro Val Gln Ala Pro Gly Gln Gln
645 650 655
Thr Phe Gln Gln Ser Ser Gly Gln Ala Pro Ser Ser Asn Ile Ser Gln
660 665 670
Gln Leu Gln Thr Ser Gln Gly Leu Gln Gln His Asp Ser His Thr Ser
675 680 685
Gln Met Pro Gln Ala Ser Leu Pro Ser Met Asn Thr Gly Val Gln Thr
690 695 700
Ser Gly Ala Pro Ala Pro Gln Gly Thr Asn Phe Gly Val Pro Thr Thr
705 710 715 720
Gln Gln Asn Val Thr Asn Ala Pro Gln Ala Gly Ser Asn Leu Glu Asn
725 730 735
Ala Gln Gly Asn Asn Phe Asn His Val Gln His Gly Ser Met Gly Ala
740 745 750
Ala Leu Gln Gln Glu Arg Thr Gly Pro Met Gln Gly Ala Leu Asn Ala
755 760 765
Gln Gln Gln Ser Ser Ser Asn Met Ile Ser Asn Asn Ala Met Ser Thr
770 775 780
Met Gln Thr Asn Asn Asn Ala Asn Ala Asn Ser Leu Gln Gln Leu Lys
785 790 795 800
Gln Gln Arg Gln Glu His Gln Met Met Gln Ser Gln Gln Met Lys Gln
805 810 815
Arg Gln Gln Met Met Gln Gln Ile Gln Gln Lys Gln Thr Leu Gln Pro
820 825 830
Gln Leu Pro Ile Gln Gln Leu Lys Lys Gln Gln Gln Gln Gly Gln Met
835 840 845
Gln Phe Pro Gln Leu His Ser Gly Asn Asp Val Asn Glu Leu Lys Val
850 855 860
Arg Gln Gly Ala Ala Ile Lys Ser Gly Met Tyr Gln Gln Leu Ser Gln
865 870 875 880
Arg Asn Tyr Tyr Gln Gln Ile Lys Gln Gly Gly Val Phe Pro Ile Ser
885 890 895
Ser Pro Gln Thr Leu Gln Thr Ser Ser Pro Gln Ile Ser His His Ser
900 905 910
Pro Gln Val Asp Gln His Ser Leu Leu Gln Ser Gln Val Lys Thr Gly
915 920 925
Thr Pro Leu His Ser Ala Asn Ser Pro Phe Val Pro Ser Pro Ser Pro
930 935 940
Pro Val Ala Pro Ser Pro Met Pro Met Asp Ser Asp Lys Pro Leu Ser
945 950 955 960
Asn Leu Ser Ser Val Thr Ser Ala Gly Gln Ala Gly His Gln Gln Thr
965 970 975
Ser Leu Ala Pro Gln Thr Gln Ser Ile Ala Val Asn Thr Pro Gly Ile
980 985 990
Ser Ala Ser Pro Leu Leu Ala Glu Phe Thr Ser Ala Asp Gly Ser Pro
995 1000 1005
Ala Asn Val Pro Thr Gln Val Pro Ala Lys Ser Ser Ala Ala Glu
1010 1015 1020
Arg Pro Leu Asp Arg Leu Leu Lys Ala Leu Arg Thr Thr Gln Arg
1025 1030 1035
Glu Ser Leu Ser Ala Ala Val Ser Asp Ile Gly Ser Val Val Ser
1040 1045 1050
Met Ile Asp Arg Ile Ala Gly Ser Ala Pro Gly Asn Gly Ser Arg
1055 1060 1065
Ala Ala Val Gly Glu Asp Leu Val Ala Met Thr Lys Cys Arg Leu
1070 1075 1080
Gln Ala Arg Asn Phe Ile Thr His Asp Gly Ser Gly Ala Ser Lys
1085 1090 1095
Lys Met Lys Arg Asp Thr Ser Ala Met Pro Leu Asn Val Ser Ser
1100 1105 1110
Ala Gly Ser Val Asn Asp Ser Leu Lys Gln Ser Tyr Gly Val Gly
1115 1120 1125
Thr Pro Glu Leu Gln Ser Thr Ala Thr Ser Arg Val Lys Trp Gln
1130 1135 1140
Arg Ala Glu Val Asn His Ala Leu Met Glu Glu Ile Gln Glu Ile
1145 1150 1155
Asn Gln Gln Leu Ile Asp Thr Glu Leu His Val Ser Glu Asp Asp
1160 1165 1170
Ala Glu Ser Phe Thr Thr Ser Glu Gly Gly Lys Gly Thr Val Ile
1175 1180 1185
Arg Cys Thr Phe Thr Ala Val Ala Val Cys Pro Ser Leu Lys Ser
1190 1195 1200
Val Phe Ala Ser Ala Gln Met Ser Pro Ile Leu Pro Leu Arg Leu
1205 1210 1215
Leu Val Pro Ala Ser Tyr Pro Lys Cys Ser Pro Val Leu Leu Asp
1220 1225 1230
Lys Phe Pro Asp Glu Gln Cys Arg Asn Ser Asp Asp Leu Ser Thr
1235 1240 1245
Lys Ala Lys Thr Lys Phe Ser Val Leu Leu Arg Gly Leu Ala Glu
1250 1255 1260
Pro Met Ser Leu Arg Glu Ile Ala Arg Thr Trp Asp Ala Cys Ala
1265 1270 1275
Arg Lys Val Ile Thr Glu Tyr Ala Gln Gln Thr Gly Gly Gly Ser
1280 1285 1290
Phe Ser Ser Ser Tyr Gly Cys Trp Glu Ser Cys Val Gly Ala Cys
1295 1300 1305
<210> 297
<211> 747
<212> ДНК
<213> Arabidopsis thaliana
<400> 297
atggcgacga ccttaagcag agatcaatat gtctacatgg cgaagctcgc cgagcaagcc
60
gagcgttacg aagagatggt tcaattcatg gaacagctcg taagtggagc tacaccggcc
120
ggtgagctga ccgtagaaga gaggaatctt ctctcggtcg cgtataagaa cgtgattgga
180
tctcttcgtg cggcatggag aatcgtgtct tcgattgagc aaaaggaaga gagcaggaag
240
aacgaagaac acgtgtcgct tgttaaggat tacagatcta aagttgagac tgagctttct
300
tcgatctgtt ctgggattct caggttactt gattcgcatc taattccttc agctactgcc
360
agtgagtcta aggtttttta cctgaagatg aaaggagatt atcatcgtta tttggctgag
420
tttaaatctg gtgatgagag gaaaactgct gctgaagata ctatgatcgc ttacaaagct
480
gctcaggacg ttgcagttgc tgatctagca cctacacatc cgatcaggct tggtttggct
540
cttaacttct cagtgtttta ctacgagatt ctcaactctt cagagaaagc ttgtagcatg
600
gcgaaacagg cttttgaaga agccattgct gagctggaca cattgggaga ggagtcatac
660
aaggacagta ctctcatcat gcagttgcta agggacaatc taaccctttg gacctccgat
720
atgcaggagc agatggatga ggcctga
747
<210> 298
<211> 248
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 298
Met Ala Thr Thr Leu Ser Arg Asp Gln Tyr Val Tyr Met Ala Lys Leu
1 5 10 15
Ala Glu Gln Ala Glu Arg Tyr Glu Glu Met Val Gln Phe Met Glu Gln
20 25 30
Leu Val Ser Gly Ala Thr Pro Ala Gly Glu Leu Thr Val Glu Glu Arg
35 40 45
Asn Leu Leu Ser Val Ala Tyr Lys Asn Val Ile Gly Ser Leu Arg Ala
50 55 60
Ala Trp Arg Ile Val Ser Ser Ile Glu Gln Lys Glu Glu Ser Arg Lys
65 70 75 80
Asn Glu Glu His Val Ser Leu Val Lys Asp Tyr Arg Ser Lys Val Glu
85 90 95
Thr Glu Leu Ser Ser Ile Cys Ser Gly Ile Leu Arg Leu Leu Asp Ser
100 105 110
His Leu Ile Pro Ser Ala Thr Ala Ser Glu Ser Lys Val Phe Tyr Leu
115 120 125
Lys Met Lys Gly Asp Tyr His Arg Tyr Leu Ala Glu Phe Lys Ser Gly
130 135 140
Asp Glu Arg Lys Thr Ala Ala Glu Asp Thr Met Ile Ala Tyr Lys Ala
145 150 155 160
Ala Gln Asp Val Ala Val Ala Asp Leu Ala Pro Thr His Pro Ile Arg
165 170 175
Leu Gly Leu Ala Leu Asn Phe Ser Val Phe Tyr Tyr Glu Ile Leu Asn
180 185 190
Ser Ser Glu Lys Ala Cys Ser Met Ala Lys Gln Ala Phe Glu Glu Ala
195 200 205
Ile Ala Glu Leu Asp Thr Leu Gly Glu Glu Ser Tyr Lys Asp Ser Thr
210 215 220
Leu Ile Met Gln Leu Leu Arg Asp Asn Leu Thr Leu Trp Thr Ser Asp
225 230 235 240
Met Gln Glu Gln Met Asp Glu Ala
245
<210> 299
<211> 762
<212> ДНК
<213> Sorghum bicolor
<400> 299
atgtcgcggg aggagaatgt ctacatggcc aagctggctg agcaagcaga acggtatgag
60
gaaatggttg agtacatgga gaaggtggct aagaccgttg atgtggaaga gctcaccgtt
120
gaggagcgta acctcctatc tgttgcctac aagaatgtga ttggggcccg ccgtgcctca
180
tggcggattg tctcctccat cgagcagaag gaggagtccc gcaagaatga ggaacatgtt
240
gctcagatta aggagtaccg tggcaagatt gaggctgaat tgagcaacat ttgtgatggt
300
atcctgaagc tgcttgactc gcaccttgtg ccttcctcca ctgctgcaga gtcgaaggtg
360
ttttacctca agatgaaggg tgattatcac aggtaccttg ctgagtttaa gactggtgct
420
gagaggaagg aatctgcaga gagcacaatg gtagcttata aggcagccca ggatattgca
480
ttggctgaac tggcacctac tcatcccata aggcttggac ttgcacttaa cttctcggtg
540
ttctattatg agattctgaa ctctccggac aaagcttgca accttgcaaa gcaggcattt
600
gatgaggcta tttctgagtt ggacacgctt ggtgaggaat cttacaaaga tagcaccttg
660
attatgcagc tcctaaggga caacttgacc ctctggacct ctgacatcac ggaggagggc
720
actgaggagg gcaaagaagc ctcgaagggt gatgctgagt ag
762
<210> 300
<211> 253
<212> БЕЛОК
<213> Sorghum bicolor
<400> 300
Met Ser Arg Glu Glu Asn Val Tyr Met Ala Lys Leu Ala Glu Gln Ala
1 5 10 15
Glu Arg Tyr Glu Glu Met Val Glu Tyr Met Glu Lys Val Ala Lys Thr
20 25 30
Val Asp Val Glu Glu Leu Thr Val Glu Glu Arg Asn Leu Leu Ser Val
35 40 45
Ala Tyr Lys Asn Val Ile Gly Ala Arg Arg Ala Ser Trp Arg Ile Val
50 55 60
Ser Ser Ile Glu Gln Lys Glu Glu Ser Arg Lys Asn Glu Glu His Val
65 70 75 80
Ala Gln Ile Lys Glu Tyr Arg Gly Lys Ile Glu Ala Glu Leu Ser Asn
85 90 95
Ile Cys Asp Gly Ile Leu Lys Leu Leu Asp Ser His Leu Val Pro Ser
100 105 110
Ser Thr Ala Ala Glu Ser Lys Val Phe Tyr Leu Lys Met Lys Gly Asp
115 120 125
Tyr His Arg Tyr Leu Ala Glu Phe Lys Thr Gly Ala Glu Arg Lys Glu
130 135 140
Ser Ala Glu Ser Thr Met Val Ala Tyr Lys Ala Ala Gln Asp Ile Ala
145 150 155 160
Leu Ala Glu Leu Ala Pro Thr His Pro Ile Arg Leu Gly Leu Ala Leu
165 170 175
Asn Phe Ser Val Phe Tyr Tyr Glu Ile Leu Asn Ser Pro Asp Lys Ala
180 185 190
Cys Asn Leu Ala Lys Gln Ala Phe Asp Glu Ala Ile Ser Glu Leu Asp
195 200 205
Thr Leu Gly Glu Glu Ser Tyr Lys Asp Ser Thr Leu Ile Met Gln Leu
210 215 220
Leu Arg Asp Asn Leu Thr Leu Trp Thr Ser Asp Ile Thr Glu Glu Gly
225 230 235 240
Thr Glu Glu Gly Lys Glu Ala Ser Lys Gly Asp Ala Glu
245 250
<210> 301
<211> 777
<212> ДНК
<213> Arabidopsis thaliana
<400> 301
atggcggcga cattaggcag agaccagtat gtgtacatgg cgaagctcgc cgagcaggcg
60
gagcgttacg aagagatggt tcaattcatg gaacagctcg ttacaggcgc tactccagcg
120
gaagagctca ccgttgaaga gaggaatctc ctctctgttg cttacaaaaa cgtgatcgga
180
tctctacgcg ccgcctggag gatcgtgtct tcgattgagc agaaggaaga gagtaggaag
240
aacgacgagc acgtgtcgct tgtcaaggat tacagatcta aagttgagtc tgagctttct
300
tctgtttgct ctggaatcct taagctcctt gactcgcatc tgatcccatc tgctggagcg
360
agtgagtcta aggtctttta cttgaagatg aaaggtgatt atcatcggta catggctgag
420
tttaagtctg gtgatgagag gaaaactgct gctgaagata ccatgctcgc ttacaaagca
480
gctcaggata tcgcagctgc ggatatggca cctactcatc cgataaggct tggtctggcc
540
ctgaatttct cagtgttcta ctatgagatt ctcaattctt cagacaaagc ttgtaacatg
600
gccaaacagg cttttgagga ggccatagct gagcttgaca ctctgggaga ggaatcctac
660
aaagacagca ctctcataat gcagttgctg agggacaatt taaccctttg gacctccgat
720
atgcaggtat tcacatctct ttatgccgta cctcgatatt ctacaccatt cgaataa
777
<210> 302
<211> 258
<212> БЕЛОК
<213> Arabidopsis thaliana
<400> 302
Met Ala Ala Thr Leu Gly Arg Asp Gln Tyr Val Tyr Met Ala Lys Leu
1 5 10 15
Ala Glu Gln Ala Glu Arg Tyr Glu Glu Met Val Gln Phe Met Glu Gln
20 25 30
Leu Val Thr Gly Ala Thr Pro Ala Glu Glu Leu Thr Val Glu Glu Arg
35 40 45
Asn Leu Leu Ser Val Ala Tyr Lys Asn Val Ile Gly Ser Leu Arg Ala
50 55 60
Ala Trp Arg Ile Val Ser Ser Ile Glu Gln Lys Glu Glu Ser Arg Lys
65 70 75 80
Asn Asp Glu His Val Ser Leu Val Lys Asp Tyr Arg Ser Lys Val Glu
85 90 95
Ser Glu Leu Ser Ser Val Cys Ser Gly Ile Leu Lys Leu Leu Asp Ser
100 105 110
His Leu Ile Pro Ser Ala Gly Ala Ser Glu Ser Lys Val Phe Tyr Leu
115 120 125
Lys Met Lys Gly Asp Tyr His Arg Tyr Met Ala Glu Phe Lys Ser Gly
130 135 140
Asp Glu Arg Lys Thr Ala Ala Glu Asp Thr Met Leu Ala Tyr Lys Ala
145 150 155 160
Ala Gln Asp Ile Ala Ala Ala Asp Met Ala Pro Thr His Pro Ile Arg
165 170 175
Leu Gly Leu Ala Leu Asn Phe Ser Val Phe Tyr Tyr Glu Ile Leu Asn
180 185 190
Ser Ser Asp Lys Ala Cys Asn Met Ala Lys Gln Ala Phe Glu Glu Ala
195 200 205
Ile Ala Glu Leu Asp Thr Leu Gly Glu Glu Ser Tyr Lys Asp Ser Thr
210 215 220
Leu Ile Met Gln Leu Leu Arg Asp Asn Leu Thr Leu Trp Thr Ser Asp
225 230 235 240
Met Gln Val Phe Thr Ser Leu Tyr Ala Val Pro Arg Tyr Ser Thr Pro
245 250 255
Phe Glu
<210> 303
<211> 762
<212> ДНК
<213> Sorghum bicolor
<400> 303
atgtcgcggg aggagaatgt ctacatggcc aagctggctg agcaagcaga acggtatgag
60
gaaatggttg agtacatgga gaaggtggct aagaccgttg atgtggaaga gctcaccgtt
120
gaggagcgta acctcctatc tgttgcctac aagaatgtga ttggggcccg ccgtgcctca
180
tggcggattg tctcctccat cgagcagaag gaggagtccc gcaagaatga ggaacatgtt
240
gctcagatta aggagtaccg tggcaagatt gaggctgaat tgagcaacat ttgtgatggt
300
atcctgaagc tgcttgactc gcaccttgtg ccttcctcca ctgctgcaga gtcgaaggtg
360
ttttacctca agatgaaggg tgattatcac aggtaccttg ctgagtttaa gactggtgct
420
gagaggaagg aatctgcaga gagcacaatg gtagcttata aggcagccca ggatattgca
480
ttggctgaac tggcacctac tcatcccata aggcttggac ttgcacttaa cttctcggtg
540
ttctattatg agattctgaa ctctccggac aaagcttgca accttgcaaa gcaggcattt
600
gatgaggcta tttctgagtt ggacacgctt ggtgaggaat cttacaaaga tagcaccttg
660
attatgcagc tcctaaggga caacttgacc ctctggacct ctgacatcac ggaggagggc
720
actgaggagg gcaaagaagc ctcgaagggt gatgctgagt ag
762
<210> 304
<211> 253
<212> БЕЛОК
<213> Sorghum bicolor
<400> 304
Met Ser Arg Glu Glu Asn Val Tyr Met Ala Lys Leu Ala Glu Gln Ala
1 5 10 15
Glu Arg Tyr Glu Glu Met Val Glu Tyr Met Glu Lys Val Ala Lys Thr
20 25 30
Val Asp Val Glu Glu Leu Thr Val Glu Glu Arg Asn Leu Leu Ser Val
35 40 45
Ala Tyr Lys Asn Val Ile Gly Ala Arg Arg Ala Ser Trp Arg Ile Val
50 55 60
Ser Ser Ile Glu Gln Lys Glu Glu Ser Arg Lys Asn Glu Glu His Val
65 70 75 80
Ala Gln Ile Lys Glu Tyr Arg Gly Lys Ile Glu Ala Glu Leu Ser Asn
85 90 95
Ile Cys Asp Gly Ile Leu Lys Leu Leu Asp Ser His Leu Val Pro Ser
100 105 110
Ser Thr Ala Ala Glu Ser Lys Val Phe Tyr Leu Lys Met Lys Gly Asp
115 120 125
Tyr His Arg Tyr Leu Ala Glu Phe Lys Thr Gly Ala Glu Arg Lys Glu
130 135 140
Ser Ala Glu Ser Thr Met Val Ala Tyr Lys Ala Ala Gln Asp Ile Ala
145 150 155 160
Leu Ala Glu Leu Ala Pro Thr His Pro Ile Arg Leu Gly Leu Ala Leu
165 170 175
Asn Phe Ser Val Phe Tyr Tyr Glu Ile Leu Asn Ser Pro Asp Lys Ala
180 185 190
Cys Asn Leu Ala Lys Gln Ala Phe Asp Glu Ala Ile Ser Glu Leu Asp
195 200 205
Thr Leu Gly Glu Glu Ser Tyr Lys Asp Ser Thr Leu Ile Met Gln Leu
210 215 220
Leu Arg Asp Asn Leu Thr Leu Trp Thr Ser Asp Ile Thr Glu Glu Gly
225 230 235 240
Thr Glu Glu Gly Lys Glu Ala Ser Lys Gly Asp Ala Glu
245 250
<210> 305
<211> 145
<212> БЕЛОК
<213> Sesamum indicum
<400> 305
Met Ala Glu His Tyr Gly Gln Gln Gln Gln Thr Arg Ala Pro His Leu
1 5 10 15
Gln Leu Gln Pro Arg Ala Gln Arg Val Val Lys Ala Ala Thr Ala Val
20 25 30
Thr Ala Gly Gly Ser Leu Leu Val Leu Ser Gly Leu Thr Leu Ala Gly
35 40 45
Thr Val Ile Ala Leu Thr Ile Ala Thr Pro Leu Leu Val Ile Phe Ser
50 55 60
Pro Val Leu Val Pro Ala Val Ile Thr Ile Phe Leu Leu Gly Ala Gly
65 70 75 80
Phe Leu Ala Ser Gly Gly Phe Gly Val Ala Ala Leu Ser Val Leu Ser
85 90 95
Trp Ile Tyr Arg Tyr Leu Thr Gly Lys His Pro Pro Gly Ala Asp Gln
100 105 110
Leu Glu Ser Ala Lys Thr Lys Leu Ala Ser Lys Ala Arg Glu Met Lys
115 120 125
Asp Arg Ala Glu Gln Phe Ser Gln Gln Pro Val Ala Gly Ser Gln Thr
130 135 140
Ser
145
<210> 306
<211> 153
<212> БЕЛОК
<213> Ficus pumila
<400> 306
Met Ala Glu Pro Gln Ser Leu Gln Arg Gly Glu Arg Gly Glu Gln Leu
1 5 10 15
Gln Leu Gln Leu Gln Gln Gln Gln His Pro Arg Ser His Gln Val Val
20 25 30
Lys Ala Ala Thr Ala Val Thr Ala Gly Gly Ser Leu Leu Val Leu Ser
35 40 45
Ala Leu Ile Leu Ala Gly Thr Val Ile Ala Leu Thr Ile Ala Thr Pro
50 55 60
Leu Phe Val Ile Phe Ser Pro Val Leu Val Pro Ala Val Ile Thr Leu
65 70 75 80
Gly Leu Ile Ile Ile Gly Phe Leu Ala Ser Gly Gly Phe Gly Val Ala
85 90 95
Ala Leu Thr Val Leu Ser Trp Ile Tyr Arg Tyr Val Thr Gly Lys His
100 105 110
Pro Pro Gly Ala Asp Gln Leu Asp Gln Ala Arg His Lys Leu Ala Ser
115 120 125
Lys Ala Arg Glu Met Lys Asp Lys Ala Glu Gln Phe Gly Gln Gln His
130 135 140
Leu Thr Ser Gly Gln Gln Gln Ser Ser
145 150
<210> 307
<211> 142
<212> БЕЛОК
<213> Cucumis sativus
<400> 307
Met Ala Glu His Gln Pro Tyr Gln Gln Ser His Gln Gln Pro Gly Ser
1 5 10 15
Gln Pro Arg Tyr Gln Val Val Lys Ala Ala Thr Ala Ala Thr Ala Gly
20 25 30
Gly Ser Leu Leu Val Leu Ser Gly Leu Ile Leu Ala Gly Thr Val Ile
35 40 45
Ala Leu Thr Ile Ala Thr Pro Leu Leu Val Ile Phe Ser Pro Val Leu
50 55 60
Val Pro Ala Val Ile Thr Val Ser Leu Leu Ile Met Gly Phe Leu Ala
65 70 75 80
Ser Gly Gly Phe Gly Val Ala Gly Ile Thr Val Phe Ser Trp Ile Tyr
85 90 95
Arg Tyr Val Thr Gly Lys His Pro Pro Gly Ala Asp Gln Leu Asp Leu
100 105 110
Ala Arg His Lys Leu Ala Ser Lys Ala Arg Glu Met Lys Asp Arg Ala
115 120 125
Glu Gln Phe Gly Gln Gln His Thr Ser Gly Pro Gln Thr Ser
130 135 140
<210> 308
<211> 155
<212> БЕЛОК
<213> Linum usitatissimum
<400> 308
Met Asp Gln Ser His Gln Thr Tyr Ala Gly Thr Met Gln Asn Pro Ser
1 5 10 15
Tyr Gly Gly Gly Gly Thr Met His Gln Gln Gln Gln Pro Arg Ser Tyr
20 25 30
Gln Ala Val Lys Ala Ala Thr Ala Ala Thr Ala Gly Gly Ser Leu Ile
35 40 45
Val Leu Ser Gly Leu Ile Leu Thr Ala Thr Val Ile Ser Leu Ile Leu
50 55 60
Ala Thr Pro Leu Leu Val Ile Phe Ser Pro Val Leu Val Pro Ala Leu
65 70 75 80
Ile Thr Val Gly Leu Leu Ile Thr Gly Phe Leu Ala Ser Gly Gly Phe
85 90 95
Gly Val Ala Ala Val Thr Val Leu Ser Trp Ile Tyr Arg Tyr Val Thr
100 105 110
Gly Gly His Pro Val Gly Ala Asp Ser Leu Glu Gln Ala Arg Ser Arg
115 120 125
Leu Ala Gly Lys Ala Arg Glu Val Lys Asp Arg Ala Ser Glu Phe Gly
130 135 140
Gln Gln His Val Thr Gly Gly Gln Gln Thr Ser
145 150 155
<210> 309
<211> 212
<212> БЕЛОК
<213> Glycine max
<400> 309
Met Gly Phe Ala Cys Lys Pro Cys Met Lys Ser Leu Pro Thr Arg Ala
1 5 10 15
Lys Pro Pro Pro Gln Val Leu Pro Pro Lys Pro Pro Leu Thr Asn Phe
20 25 30
Ser Ile Tyr Thr Leu Ile Thr Thr Thr Leu Asn Pro Thr Thr Leu Ile
35 40 45
Thr His Ser Ser Leu Lys Thr Leu Val Phe His Phe Asp Asn Ser His
50 55 60
Thr Met Ala Glu Leu His Tyr Gln Gln Gln His Gln Tyr Pro His Arg
65 70 75 80
Tyr Pro Asn Asp Pro Tyr Glu Gln Gln Thr Ser Tyr Ser Thr Gln Val
85 90 95
Val Lys Ala Ala Thr Ala Val Thr Ala Gly Gly Ser Leu Leu Ile Leu
100 105 110
Ala Ser Leu Ile Leu Ala Gly Thr Ile Ile Ala Leu Thr Ile Val Thr
115 120 125
Pro Pro Leu Val Ile Phe Ser Pro Val Leu Val Pro Ala Val Ile Thr
130 135 140
Val Ala Leu Leu Ser Leu Gly Phe Leu Ala Ser Gly Gly Phe Gly Val
145 150 155 160
Ala Ala Ile Thr Val Leu Ala Trp Ile Tyr Arg Tyr Val Thr Gly Lys
165 170 175
Tyr Pro Pro Gly Ala Asp Gln Leu Asp Ser Ala Pro His Lys Ile Met
180 185 190
Asp Lys Ala Arg Glu Ile Lys Asp Tyr Gly Gln Gln Gln Ile Ser Gly
195 200 205
Val Gln Ala Ser
210
<210> 310
<211> 149
<212> БЕЛОК
<213> Ananas comosus
<400> 310
Met Ala Asp Tyr Gln Arg Glu Gln Arg Gly Gly Gly Phe Met Gln Gly
1 5 10 15
Gln Gln Ser Gln Gln Gln Gln Gln Gln Gln Gln Pro Met Met Met Thr
20 25 30
Ala Val Lys Ala Ala Thr Ala Ala Thr Ala Gly Gly Ser Met Leu Val
35 40 45
Leu Ser Gly Leu Thr Leu Ala Gly Thr Val Ile Ala Leu Thr Val Ala
50 55 60
Thr Pro Leu Leu Val Ile Phe Ser Pro Val Leu Val Pro Ala Thr Ile
65 70 75 80
Ala Val Ser Leu Leu Ala Ala Gly Phe Val Thr Ser Gly Gly Leu Gly
85 90 95
Leu Ala Ala Leu Ser Val Leu Ser Trp Met Tyr Lys Tyr Leu Thr Gly
100 105 110
Lys His Pro Pro Gly Ala Asp Gln Leu Glu His Ala Lys Ala Arg Leu
115 120 125
Ala Ser Lys Ala Arg Asp Ile Lys Glu Ser Ala Gln His Arg Ile Asp
130 135 140
Gln Ala Gln Gly Ser
145
<210> 311
<211> 154
<212> БЕЛОК
<213> Setaria italica
<400> 311
Met Ala Asp Gln His Arg Gly Ala Met Gly Gly Gly Gly Gly Gly Tyr
1 5 10 15
Gly Asp Leu His Arg Gly Gly Glu Arg Gly Glu Thr Gln Gln Arg Gln
20 25 30
Ser Ala Met Met Thr Ala Leu Lys Ala Ala Thr Ala Ala Thr Ala Gly
35 40 45
Gly Ser Met Leu Val Leu Ser Gly Leu Ile Leu Ala Gly Thr Val Ile
50 55 60
Ala Leu Thr Val Ala Thr Pro Val Leu Val Ile Phe Ser Pro Val Leu
65 70 75 80
Val Pro Ala Ala Ile Thr Leu Ala Leu Met Ala Ala Gly Phe Val Thr
85 90 95
Ser Gly Gly Leu Gly Val Ala Ala Leu Ser Val Phe Ser Trp Met Tyr
100 105 110
Lys Tyr Leu Thr Gly Lys His Pro Pro Gly Ala Asp Gln Leu Asp His
115 120 125
Ala Lys Ala Arg Leu Ala Ser Lys Ala Arg Asp Ile Lys Asp Ala Ala
130 135 140
Gln His Arg Ile Asp Gln Ala Gln Gly Ser
145 150
<210> 312
<211> 157
<212> БЕЛОК
<213> Fragaria vesca
<400> 312
Met Ala Glu Leu His Gln Gln Tyr Gln Gln Ser His Pro Phe Gln Ala
1 5 10 15
Gln Gln Gly Met Met Gln Gln Gln Pro Lys Ser Tyr Gln Val Ala Lys
20 25 30
Ala Ala Thr Ala Val Thr Ala Gly Gly Ser Leu Leu Val Leu Ser Gly
35 40 45
Leu Val Leu Ala Gly Thr Val Ile Cys Leu Thr Val Ala Thr Pro Leu
50 55 60
Leu Val Ile Phe Ser Pro Val Leu Val Pro Ala Val Ile Thr Val Ala
65 70 75 80
Leu Ile Met Thr Gly Phe Leu Ala Ser Gly Gly Phe Gly Val Ala Ala
85 90 95
Ile Ser Val Leu Ser Trp Ile Tyr Lys Tyr Val Thr Gly Ser His Pro
100 105 110
Pro Gly Ala Asp Lys Leu Asp Ser Ala Arg His Lys Leu Ala Gly Lys
115 120 125
Ala Arg Asp Met Lys Asp Arg Ala Glu Gln Phe Gly Gln Gln His Met
130 135 140
Gly Thr Glu Arg Gly Gln His Gly Gln His Gln Thr Ser
145 150 155
<210> 313
<211> 170
<212> БЕЛОК
<213> Brassica napus
<400> 313
Met Thr Asp Thr Ala Arg Thr His His Asp Val Thr Asn Arg Asp Gln
1 5 10 15
Tyr Ser Met Met Gly Arg Asp Arg Asp Gln Tyr Asn Met Tyr Gly Arg
20 25 30
Asp Tyr Ser Lys Ser Arg Gln Ile Ala Lys Ala Val Thr Ala Val Thr
35 40 45
Ala Gly Gly Ser Leu Leu Val Leu Ser Ser Leu Thr Leu Val Gly Thr
50 55 60
Val Ile Ala Leu Thr Val Ala Thr Pro Leu Leu Val Ile Phe Ser Pro
65 70 75 80
Ile Leu Val Pro Ala Leu Ile Thr Val Ala Met Leu Ile Thr Gly Phe
85 90 95
Leu Ser Ser Gly Gly Phe Gly Ile Ala Ala Ile Thr Val Phe Ser Trp
100 105 110
Ile Tyr Lys Tyr Ala Thr Gly Glu His Pro Gln Gly Ser Asp Lys Leu
115 120 125
Asp Ser Ala Arg Met Lys Leu Gly Ser Lys Ala Gln Asp Leu Lys Asp
130 135 140
Arg Ala Gln Tyr Tyr Gly Gln Gln His Thr Gly Gly Glu His Asp Arg
145 150 155 160
Asp Arg Thr Arg Gly Thr Gln His Thr Thr
165 170
<210> 314
<211> 147
<212> БЕЛОК
<213> Solanum lycopersicum
<400> 314
Met Ala Asp Tyr Tyr Gly Gln Pro Gln His Thr Gln His Gln Phe Phe
1 5 10 15
His Gly Gly Gln Gln Pro Arg Ser His Gln Met Val Lys Ala Ala Thr
20 25 30
Ala Val Thr Ala Gly Gly Ser Leu Leu Leu Leu Ser Gly Leu Thr Leu
35 40 45
Ala Ala Thr Val Ile Ala Leu Thr Ile Ala Thr Pro Val Leu Val Ile
50 55 60
Phe Ser Pro Val Ile Val Pro Ala Val Ile Thr Leu Phe Leu Leu Phe
65 70 75 80
Ser Gly Phe Leu Ala Ser Gly Gly Phe Gly Val Ala Ala Val Ser Val
85 90 95
Leu Ser Trp Ile Tyr Arg Tyr Val Thr Gly Lys Arg Pro Pro Gly Ala
100 105 110
Asp Gln Leu Glu Gln Ala Arg His Lys Leu Ala Thr Lys Ala Gly Glu
115 120 125
Met Lys Asp Lys Ala Gln Glu Phe Gly Gln Gln His Ile Thr Gly Thr
130 135 140
His Gln Thr
145
<210> 315
<211> 163
<212> БЕЛОК
<213> Vanilla planifolia
<400> 315
Met Ala Asp Ser Gln Ala Gly Pro Arg Val Pro Val Val Asn Gly Asn
1 5 10 15
Gly Gly Ile Gly Gly Cys His Ala Leu Leu Arg Arg Ile His His His
20 25 30
Ser Pro Asn Ser Ser Gln Val Val Gly Phe Leu Thr Leu Ala Leu Ser
35 40 45
Gly Ala Ile Leu Leu Leu Leu Ile Gly Ile Thr Val Ile Gly Ala Thr
50 55 60
Leu Gly Phe Ile Ala Phe Val Pro Leu Leu Leu Met Thr Ser Pro Val
65 70 75 80
Trp Val Pro Leu Val Ser Ser Ala Phe Val Leu Leu Ala Gly Ala Ala
85 90 95
Ala Phe Ser Gly Thr Ala Val Ala Thr Ala Ala Gly Val Thr Trp Leu
100 105 110
Tyr Arg Tyr Phe Thr Gly Arg His Pro Val Gly Ser Asp Arg Val Asp
115 120 125
Tyr Ala Arg Ser Arg Ile Ala Asp Thr Ala Ser His Val Lys Asp Tyr
130 135 140
Ala Arg Glu Tyr Gly Gly Tyr Ile Gln Ser Arg Ile Lys Asp Ala Ala
145 150 155 160
Pro Gly Ala
<210> 316
<211> 241
<212> БЕЛОК
<213> Triadica sebifera
<400> 316
Met Ala Asp Gly Asn Val Asn Ser Gln Glu Gln Met Ala Lys Gln Glu
1 5 10 15
Glu Gln Arg Leu Lys Tyr Leu Glu Phe Val Gln Val Ala Ala Ile His
20 25 30
Ala Val Val Thr Phe Thr Asn Leu Tyr Val Tyr Ala Lys Asn Lys Ser
35 40 45
Gly Pro Leu Lys Pro Gly Val Glu Thr Val Glu Gly Thr Val Lys Ser
50 55 60
Val Val Gly Pro Val Tyr Gly Lys Phe His Asp Val Pro Ile Glu Val
65 70 75 80
Leu Lys Phe Val Asp Arg Lys Ile Asp Gln Ser Val Ser Ser Leu Asp
85 90 95
Ser Arg Val Pro Pro Val Val Lys Gln Leu Ser Ala Gln Ala Phe Ser
100 105 110
Val Ala Arg Glu Ala Pro Val Ala Ala Arg Ala Val Ala Ser Glu Val
115 120 125
Gln Thr Ala Gly Val Lys Glu Thr Ala Ser Gly Leu Ala Arg Thr Leu
130 135 140
Tyr Phe Lys Tyr Glu Pro Lys Ala Lys Glu Leu Tyr Thr Lys Tyr Glu
145 150 155 160
Pro Lys Ala Glu Gln Cys Ala Ala Ser Ala Trp Arg Lys Leu Asn Gln
165 170 175
Leu Pro Val Phe Pro His Val Ala Gln Val Val Met Pro Thr Ala Ala
180 185 190
Tyr Cys Ser Glu Lys Tyr Asn Gln Ala Val Leu Thr Thr Ala Glu Lys
195 200 205
Gly Tyr Arg Val Ser Ser Tyr Leu Pro Phe Val Pro Thr Glu Arg Ile
210 215 220
Ala Lys Leu Phe Arg Asn Glu Ala Pro Glu Ser Thr Pro Phe Leu Ser
225 230 235 240
Asn
<210> 317
<211> 225
<212> БЕЛОК
<213> Triadica sebifera
<400> 317
Met Glu Thr Glu Lys Lys Ile Pro Glu Leu Lys His Leu Gly Phe Val
1 5 10 15
Arg Met Ala Ala Ile Gln Ser Leu Ile Cys Val Ser Asn Leu Tyr Asp
20 25 30
Tyr Ala Lys His Asn Ser Gly Pro Leu Arg Ser Thr Val Gly Thr Val
35 40 45
Glu Gly Ala Val Thr Thr Val Val Gly Pro Val Tyr Gln Lys Phe Lys
50 55 60
Asp Leu Pro Asp Asp Val Leu Val Tyr Val Asp Lys Lys Val Asp Glu
65 70 75 80
Gly Thr His Lys Phe Asp Lys His Ala Pro Pro Ile Ala Lys Lys Ala
85 90 95
Ala Ser Gln Ala His Ser Leu Phe His Ile Ala Leu Glu Lys Val Glu
100 105 110
Lys Leu Val Gln Glu Ala Arg Ala Gly Gly Pro Arg Ala Ala Leu His
115 120 125
Tyr Val Ala Thr Glu Ser Lys His Leu Ala Leu Thr Gln Ser Val Lys
130 135 140
Leu Tyr Ile Lys Leu Asn Gln Phe Pro Val Ile His Thr Val Thr Asp
145 150 155 160
Val Thr Leu Pro Thr Ala Thr His Trp Ser Asp Lys Tyr Asn His Thr
165 170 175
Val Met Asp Leu Thr Arg Lys Gly Tyr Thr Ile Phe Gly Tyr Leu Pro
180 185 190
Leu Val Pro Ile Asp Asp Ile Ser Lys Thr Phe Lys Gln Ser Lys Ala
195 200 205
Glu Glu Lys Glu Lys Ala Thr Thr His Lys Ser Asp Ser Ser Asp Ser
210 215 220
Asp
225
<210> 318
<211> 236
<212> БЕЛОК
<213> Triadica sebifera
<400> 318
Met Ala Glu Ser Glu Leu Asn Gln His Thr Asp Met Val Gln Asp Asp
1 5 10 15
Asp Lys Lys Leu Lys Tyr Leu Asp Phe Val Gln Val Ala Ala Ile Tyr
20 25 30
Val Val Val Cys Phe Ser Ser Ile Tyr Glu Tyr Ala Lys Glu Asn Ser
35 40 45
Gly Pro Leu Lys Pro Gly Val Gln Ala Val Glu Cys Thr Val Lys Thr
50 55 60
Val Ile Ser Pro Val Tyr Glu Lys Phe Arg Asp Val Pro Phe Glu Leu
65 70 75 80
Leu Lys Phe Val Asp Arg Lys Val Asp Asn Ser Leu Gly Glu Leu Asp
85 90 95
Arg His Val Pro Ser Leu Val Lys Gln Ala Ser Ser Gln Ala Arg Ala
100 105 110
Val Ala Ser Glu Ile Gln His Ala Gly Leu Val Asp Ala Thr Lys Asn
115 120 125
Ile Ala Lys Thr Met Tyr Thr Lys Tyr Glu Leu Thr Ala Trp Gln Leu
130 135 140
Tyr Cys Lys Tyr Lys Pro Val Ala Lys Arg Tyr Ala Val Ser Thr Trp
145 150 155 160
Arg Ser Leu Asn Gln Leu Pro Leu Phe Pro Gln Ala Ala Gln Ile Ala
165 170 175
Ile Pro Thr Ala Ala Ser Trp Ser Glu Lys Tyr Asn Lys Met Val Arg
180 185 190
Tyr Thr Lys Asp Arg Gly Tyr Pro Ala Ala Val Tyr Leu Pro Leu Ile
195 200 205
Ser Val Glu Arg Ile Ala Lys Val Phe Asn Glu Asp Leu Asn Gly Pro
210 215 220
Thr Val Pro Thr Asn Gly Ser Ser Ala Ala Ala Gln
225 230 235
<210> 319
<211> 371
<212> БЕЛОК
<213> Cocos nucifera
<400> 319
Met Val Glu Leu Arg Ser Ser Ser Ser Glu Met Asp Leu Asp Arg Pro
1 5 10 15
Asn Ile Glu Glu Tyr Leu Thr Thr Asp Ser Ile Gln Glu Ser Pro Lys
20 25 30
Lys Leu His Leu Arg Asp Leu Leu Asp Ile Ser Pro Thr Leu Thr Glu
35 40 45
Ala Thr Gly Ala Ile Val Asp Asp Ser Phe Thr Arg Cys Phe Lys Ser
50 55 60
Asn Pro Pro Glu Pro Trp Asn Trp Asn Val Tyr Leu Phe Pro Leu Trp
65 70 75 80
Cys Leu Gly Val Ile Ile Arg Tyr Gly Ile Leu Phe Pro Leu Arg Val
85 90 95
Ala Ile Leu Thr Ala Gly Trp Leu Val Phe Phe Ala Ala Phe Ile Pro
100 105 110
Val His Phe Leu Leu Thr Ala His Asn Lys Trp Arg Arg Lys Ile Glu
115 120 125
Arg Lys Leu Val Glu Met Ile Cys Ser Val Phe Val Ala Ser Trp Thr
130 135 140
Gly Val Val Lys Tyr His Gly Pro Arg Pro Ser Met Arg Pro Gln Gln
145 150 155 160
Val Phe Val Ala Asn His Thr Ser Met Ile Asp Phe Ile Ile Leu Glu
165 170 175
Gln Met Thr Ala Phe Ala Val Ile Met Gln Lys His Pro Gly Trp Val
180 185 190
Gly Phe Ile Gln Lys Thr Ile Leu Glu Gly Val Gly Cys Ile Trp Phe
195 200 205
Asn Arg Thr Glu Ser Lys Asp Arg Glu Val Val Ala Arg Lys Leu Arg
210 215 220
Glu His Ile His Gly Ala Asp Asn Asn Pro Leu Leu Ile Phe Pro Glu
225 230 235 240
Gly Thr Cys Val Asn Asn His Tyr Thr Val Met Phe Lys Lys Gly Ala
245 250 255
Phe Glu Leu Gly Cys Ala Val Cys Pro Val Ala Ile Lys Tyr Asn Lys
260 265 270
Ile Phe Val Asp Ala Phe Trp Asn Ser Lys Lys Gln Ser Phe Thr Met
275 280 285
His Leu Phe His Leu Met Thr Ser Trp Ala Val Val Cys Asp Val Trp
290 295 300
Tyr Leu Glu Pro Gln Tyr Ile Arg Pro Gly Glu Thr Pro Ile Glu Phe
305 310 315 320
Ala Glu Arg Val Arg Asp Met Ile Ser Val Arg Ala Gly Leu Lys Lys
325 330 335
Val Pro Trp Asp Gly Tyr Leu Lys Tyr Phe Arg Pro Ser Pro Lys Leu
340 345 350
Thr Glu Arg Lys Gln Gln Ile Phe Ala Glu Ser Val Leu Gln Arg Leu
355 360 365
Glu Glu Lys
370
<210> 320
<211> 389
<212> БЕЛОК
<213> Zea mays
<400> 320
Met Gly Tyr Ile Gly His His Gly Val Ala Thr Leu Arg Arg Tyr Lys
1 5 10 15
Tyr Ser Gly Val Asp His Ser Leu Val Ala Lys Tyr Ile Leu Gln Pro
20 25 30
Phe Trp Ser Arg Phe Val Asn Ile Phe Pro Leu Trp Phe Pro Pro Asn
35 40 45
Met Ile Thr Leu Thr Gly Phe Met Phe Leu Leu Thr Ser Ala Phe Leu
50 55 60
Gly Phe Leu Tyr Ser Pro His Leu Asp Thr Ala Pro Pro Arg Trp Val
65 70 75 80
His Leu Ala His Gly Leu Leu Leu Phe Leu Tyr Gln Thr Phe Asp Ala
85 90 95
Val Asp Gly Lys Gln Ala Arg Arg Thr Asn Ser Ser Ser Pro Leu Gly
100 105 110
Glu Leu Phe Asp His Gly Cys Asp Ala Leu Ala Cys Ala Phe Glu Ser
115 120 125
Leu Ala Phe Gly Ser Thr Ala Met Cys Gly Lys Ala Thr Phe Trp Phe
130 135 140
Trp Phe Ile Ser Ala Val Pro Phe Tyr Phe Ala Thr Trp Glu His Phe
145 150 155 160
Phe Thr Asn Thr Leu Ile Leu Pro Ile Val Asn Gly Pro Thr Glu Gly
165 170 175
Leu Met Leu Ile Tyr Leu Cys His Phe Phe Thr Phe Phe Thr Gly Ala
180 185 190
Glu Trp Trp Ala Gln Asp Phe Gln Lys Ser Met Pro Leu Leu Gly Trp
195 200 205
Val Pro Leu Ile Ser Glu Ile Pro Val Tyr Asp Ile Val Leu Cys Leu
210 215 220
Met Ile Ala Phe Ala Val Ile Pro Thr Ile Gly Ser Asn Ile His Asn
225 230 235 240
Val Tyr Lys Val Val Glu Ala Arg Lys Gly Ser Met Leu Leu Ala Leu
245 250 255
Ala Met Leu Phe Pro Phe Gly Leu Leu Leu Ala Gly Val Leu Val Trp
260 265 270
Ser Tyr Leu Ser Pro Ser Asp Ile Met Arg Thr Gln Pro His Leu Leu
275 280 285
Ile Ile Gly Thr Gly Phe Ala Phe Gly Phe Leu Val Gly Arg Met Ile
290 295 300
Leu Ala His Leu Cys Asp Glu Pro Lys Gly Leu Lys Thr Gly Met Cys
305 310 315 320
Met Ser Leu Ala Tyr Phe Pro Phe Ala Ile Ala Asn Ala Leu Thr Ala
325 330 335
Arg Leu Asp Asp Gly Asn Pro Leu Val Asp Glu Gln Leu Val Leu Leu
340 345 350
Met Tyr Cys Leu Phe Thr Val Ala Leu Tyr Met His Phe Ala Thr Ser
355 360 365
Val Ile His Glu Ile Thr Asn Ala Leu Gly Ile His Cys Phe Arg Ile
370 375 380
Thr Arg Lys Lys Ala
385
<210> 321
<211> 393
<212> БЕЛОК
<213> Zea mays
<400> 321
Met Gly Phe Ile Gly Ala His Gly Val Glu Thr Leu Lys Arg Tyr Arg
1 5 10 15
Tyr Ser Gly Glu Asp Arg Ser Val Val Ala Lys Tyr Val Leu Gln Pro
20 25 30
Phe Trp Ser Arg Cys Val Thr Leu Phe Pro Leu Trp Met Pro Pro Asn
35 40 45
Met Ile Thr Leu Thr Gly Phe Ser Phe Leu Val Leu Ser Ala Leu Leu
50 55 60
Gly Tyr Ile Cys Ser Pro His Leu Asp Thr Ala Pro Pro Arg Trp Val
65 70 75 80
His Leu Ala His Gly Leu Leu Leu Phe Leu Tyr Gln Thr Phe Asp Ala
85 90 95
Val Asp Gly Lys Gln Ala Arg Arg Thr Ser Ser Ser Ser Pro Leu Gly
100 105 110
Glu Leu Phe Asp His Gly Cys Asp Ala Leu Ala Cys Ala Phe Glu Ala
115 120 125
Leu Ala Leu Gly Ser Thr Leu Met Cys Gly Gly Trp Thr Leu Cys Phe
130 135 140
Trp Val Val Ala Ala Val Pro Phe Tyr Leu Ala Thr Trp Glu His Phe
145 150 155 160
Phe Thr Asn Thr Leu Ile Leu Pro Thr Ile Asn Gly Pro Thr Glu Gly
165 170 175
Leu Met Leu Ile Tyr Val Ser His Leu Phe Thr Phe Phe Thr Gly Ala
180 185 190
Glu Trp Trp Ala Gln Asp Phe Arg Lys Ser Leu Pro Phe Phe Gly Trp
195 200 205
Ile Pro Leu Pro Phe Leu Ser Glu Ile Glu Ile Pro Leu Tyr Val Leu
210 215 220
Val Leu Ile Leu Met Ile Val Cys Ala Val Ile Pro Thr Val Arg Ser
225 230 235 240
Asn Val Ser Asn Val Gln Glu Val Val Glu Ala Arg Lys Gly Ser Met
245 250 255
Ala Leu Ala Leu Ala Met Ile Leu Pro Phe Ile Val Leu Leu Ala Gly
260 265 270
Val Ala Ile Trp Cys Tyr Leu Ser Pro Ser Ser Ile Met Arg Asn Gln
275 280 285
Pro His Leu Leu Val Ile Gly Thr Gly Phe Asn Phe Gly Tyr Leu Val
290 295 300
Gly Arg Met Ile Leu Ala His Leu Cys Asp Glu Pro Lys Gly Leu Lys
305 310 315 320
Ser Gly Met Phe Met Ser Leu Val Phe Leu Cys Phe Pro Ile Ala Asn
325 330 335
Ala Leu Ile Ala Lys Ile Asn Asp Gly Ser Pro Leu Val Asp Glu Leu
340 345 350
Val Leu Leu Leu Leu Tyr Cys Ala Tyr Thr Val Gly Leu Tyr Leu His
355 360 365
Leu Ala Val Ser Val Val His Glu Ile Lys Asp Ala Leu Gly Ile Tyr
370 375 380
Cys Phe Arg Ile Thr Arg Lys Glu Ala
385 390
<210> 322
<211> 389
<212> БЕЛОК
<213> Sorghum bicolor
<400> 322
Met Gly Tyr Ile Gly His His Gly Val Ala Thr Leu Arg Arg Tyr Lys
1 5 10 15
Tyr Ser Gly Val Asp His Ser Leu Val Ala Lys Tyr Ile Leu Gln Pro
20 25 30
Phe Trp Ser Arg Phe Val Asn Ile Phe Pro Leu Trp Phe Pro Pro Asn
35 40 45
Met Ile Thr Leu Thr Gly Phe Met Phe Leu Leu Thr Ser Ala Phe Leu
50 55 60
Gly Phe Leu Tyr Ser Pro His Leu Asp Thr Ala Pro Pro Arg Trp Val
65 70 75 80
His Leu Ala His Gly Leu Leu Leu Phe Leu Tyr Gln Thr Phe Asp Ala
85 90 95
Val Asp Gly Lys Gln Ala Arg Arg Thr Asn Ser Ser Ser Pro Leu Gly
100 105 110
Glu Leu Phe Asp His Gly Cys Asp Ala Leu Ala Cys Ala Phe Glu Ser
115 120 125
Leu Ala Phe Gly Ser Thr Ala Met Cys Gly Lys Ala Thr Phe Trp Phe
130 135 140
Trp Phe Ile Ser Ala Val Pro Phe Tyr Phe Ala Thr Trp Glu His Phe
145 150 155 160
Phe Thr Asn Thr Leu Ile Leu Pro Ile Val Asn Gly Pro Thr Glu Gly
165 170 175
Leu Met Leu Ile Tyr Leu Cys His Phe Phe Thr Phe Phe Thr Gly Ala
180 185 190
Lys Trp Trp Ala Gln Asp Phe Gln Lys Ser Met Pro Leu Leu Gly Trp
195 200 205
Val Pro Phe Ile Ser Glu Ile Pro Val Tyr Asp Ile Val Leu Cys Leu
210 215 220
Met Ile Ala Phe Ala Val Ile Pro Thr Ile Ala Ser Asn Ile His Asn
225 230 235 240
Val Tyr Lys Val Val Glu Ala Arg Lys Gly Ser Met Val Leu Ala Leu
245 250 255
Ala Met Leu Phe Pro Phe Gly Leu Leu Leu Ala Gly Val Leu Val Trp
260 265 270
Ser Tyr Phe Ser Pro Ser Asp Ile Met Arg Asn Gln Pro His Leu Leu
275 280 285
Ile Ile Gly Thr Gly Phe Ala Phe Gly Phe Leu Val Gly Arg Met Ile
290 295 300
Leu Ala His Leu Cys Asp Glu Pro Lys Gly Leu Lys Thr Gly Met Cys
305 310 315 320
Met Ser Leu Ala Tyr Phe Pro Phe Ala Ile Ala Asn Ala Leu Thr Ala
325 330 335
Arg Leu Asp Asp Gly Asn Pro Leu Val Asp Glu Gln Leu Val Leu Leu
340 345 350
Met Tyr Cys Leu Phe Thr Val Ala Leu Tyr Met His Phe Ala Thr Ser
355 360 365
Val Ile His Glu Ile Thr Asn Ala Leu Gly Ile His Cys Phe Arg Ile
370 375 380
Thr Thr Lys Lys Ala
385
<210> 323
<211> 393
<212> БЕЛОК
<213> Sorghum bicolor
<400> 323
Met Gly Phe Ile Gly Ala His Gly Val Glu Thr Leu Lys Arg Tyr Arg
1 5 10 15
Tyr Ser Gly Glu Asp Arg Ser Val Val Ala Lys Tyr Val Leu Gln Pro
20 25 30
Phe Trp Ser Arg Cys Val Thr Leu Phe Pro Leu Trp Met Pro Pro Asn
35 40 45
Met Ile Thr Leu Thr Gly Phe Ser Phe Leu Val Leu Ser Ala Leu Leu
50 55 60
Gly Tyr Ile Tyr Ser Pro Arg Leu Asp Thr Ala Pro Pro Arg Trp Val
65 70 75 80
His Leu Ala His Gly Leu Leu Leu Phe Leu Tyr Gln Thr Phe Asp Ala
85 90 95
Val Asp Gly Lys Gln Ala Arg Arg Thr Ser Ser Ser Ser Pro Leu Gly
100 105 110
Glu Leu Phe Asp His Gly Cys Asp Ala Leu Ala Cys Ala Phe Glu Ala
115 120 125
Leu Ala Leu Gly Ser Thr Leu Met Cys Gly Gly Trp Thr Phe Trp Phe
130 135 140
Trp Val Val Ala Ala Val Pro Phe Tyr Leu Ala Thr Trp Glu His Phe
145 150 155 160
Phe Thr Asn Thr Leu Ile Leu Pro Thr Ile Asn Gly Pro Thr Glu Gly
165 170 175
Leu Met Leu Ile Tyr Val Ser His Leu Phe Thr Phe Leu Thr Gly Ala
180 185 190
Glu Trp Trp Ala Gln Asp Phe Arg Lys Ser Leu Pro Phe Leu Gly Trp
195 200 205
Ile Pro Leu Pro Phe Leu Ser Glu Ile Lys Ile Pro Leu Tyr Val Leu
210 215 220
Val Leu Ile Leu Met Ile Val Cys Ala Val Ile Pro Thr Val Arg Ser
225 230 235 240
Asn Val Ser Asn Val Gln Glu Val Val Glu Thr Arg Lys Gly Ser Met
245 250 255
Ala Leu Ala Leu Ala Met Ile Leu Pro Phe Ile Thr Leu Leu Ala Gly
260 265 270
Val Ala Ile Trp Ser Tyr Leu Ser Pro Ser Ser Ile Met Arg Asn Gln
275 280 285
Pro His Leu Leu Val Ile Gly Thr Gly Phe Asn Phe Gly Tyr Leu Val
290 295 300
Gly Arg Met Ile Leu Ala His Leu Cys Asp Glu Pro Lys Gly Leu Lys
305 310 315 320
Ser Gly Met Phe Met Ser Leu Val Phe Leu Cys Phe Pro Ile Ala Asn
325 330 335
Ala Leu Ile Ala Lys Ile Asn Asn Gly Thr Pro Leu Val Asp Glu Leu
340 345 350
Val Leu Leu Leu Leu Tyr Cys Ala Tyr Thr Val Gly Leu Tyr Leu His
355 360 365
Leu Ala Val Ser Val Val His Glu Ile Lys Asp Ala Leu Gly Ile Tyr
370 375 380
Cys Phe Arg Ile Thr Arg Lys Glu Ala
385 390
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Гибридные белки NPP1 | 2011 |
|
RU2760943C2 |
| СПОСОБЫ ПОЛУЧЕНИЯ ПРОМЫШЛЕННЫХ ПРОДУКТОВ ИЗ РАСТИТЕЛЬНЫХ ЛИПИДОВ | 2015 |
|
RU2743384C2 |
| ЭКСПРЕССИЯ ПОЛИПЕПТИДОВ НИТРОГЕНАЗЫ В РАСТИТЕЛЬНЫХ КЛЕТКАХ | 2020 |
|
RU2833880C2 |
| ГЕНЫ ТОКСИНОВ И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2014 |
|
RU2825305C2 |
| СПОСОБЫ УСОВЕРШЕНСТВОВАНИЯ ОДНОДОЛЬНЫХ РАСТЕНИЙ | 2015 |
|
RU2727428C2 |
| СПОСОБЫ УСОВЕРШЕНСТВОВАНИЯ РАСТЕНИЙ | 2015 |
|
RU2727424C2 |
| ЭКСПРЕССИЯ ПОЛИПЕПТИДОВ НИТРОГЕНАЗЫ В РАСТИТЕЛЬНЫХ КЛЕТКАХ | 2018 |
|
RU2809244C2 |
| ПЕСТИЦИДНЫЕ ГЕНЫ И СПОСОБЫ ИСПОЛЬЗОВАНИЯ | 2016 |
|
RU2746927C2 |
| ГЕНЫ ТОКСИНОВ И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2014 |
|
RU2723717C2 |
| ПЕСТИЦИДНЫЕ ГЕНЫ И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2015 |
|
RU2727665C2 |
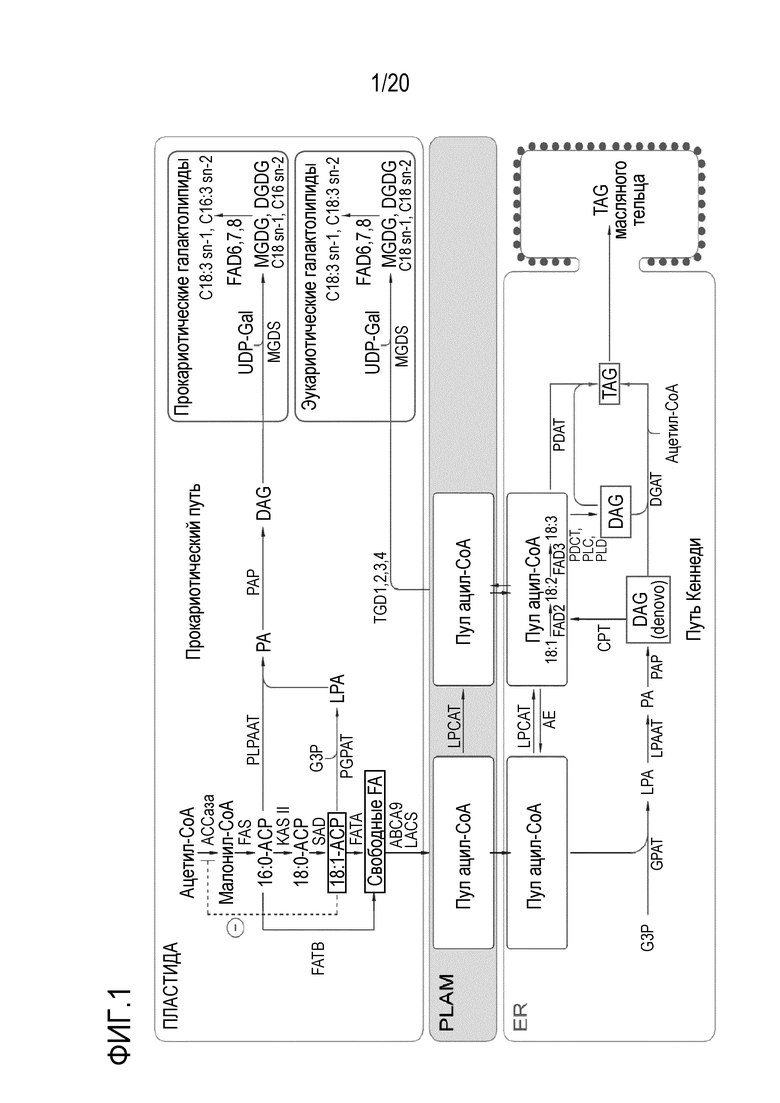

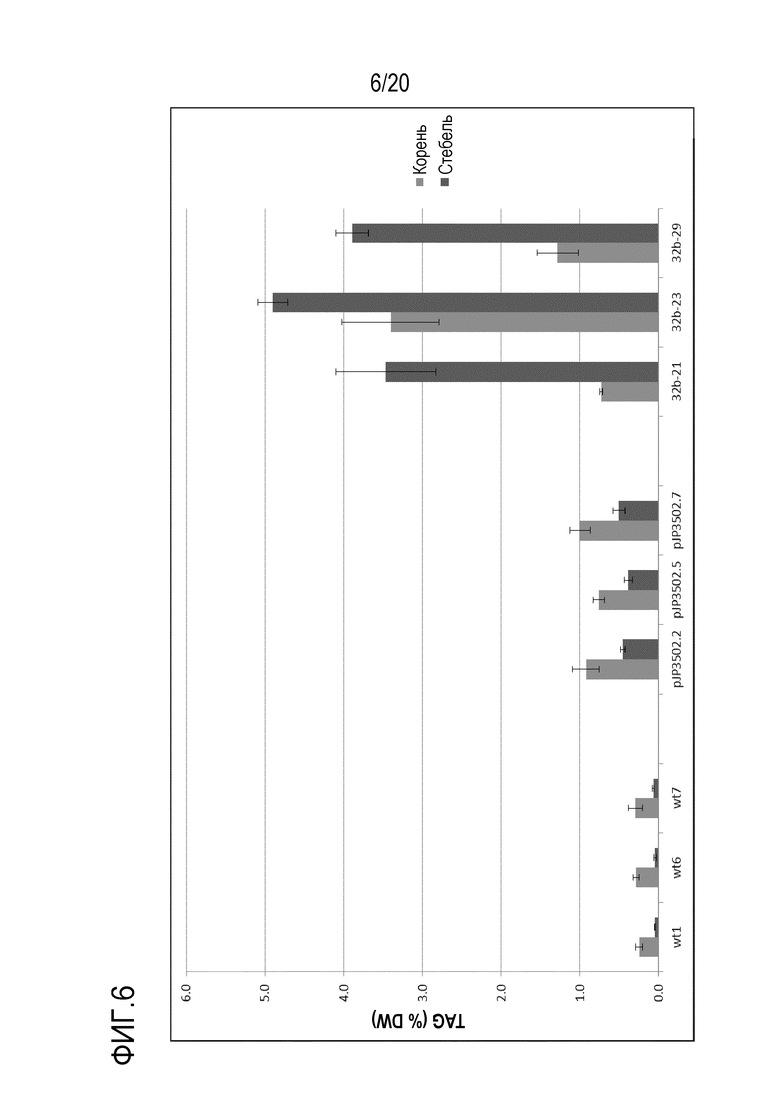
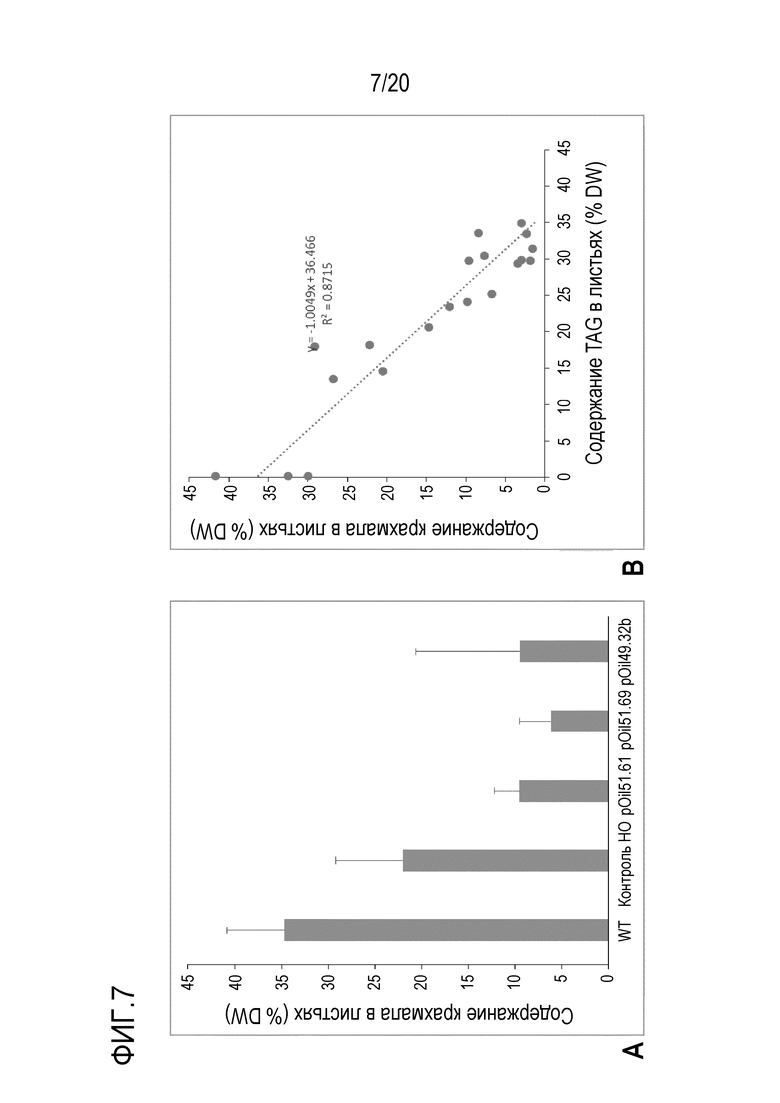
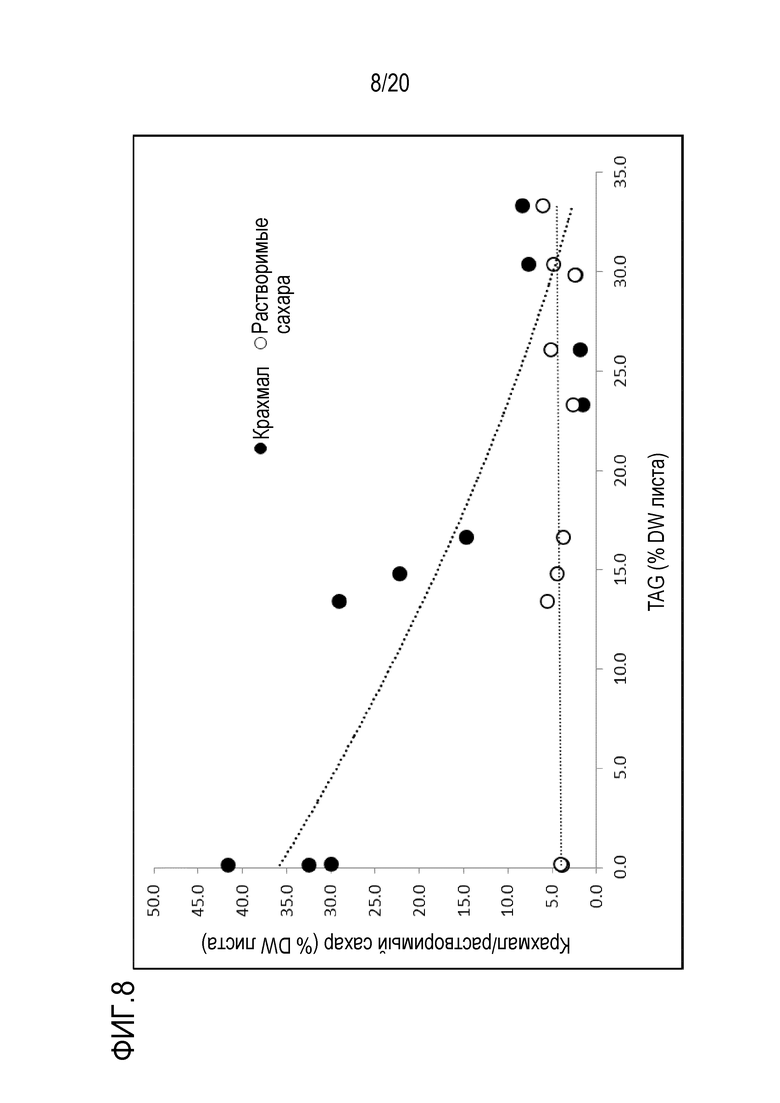
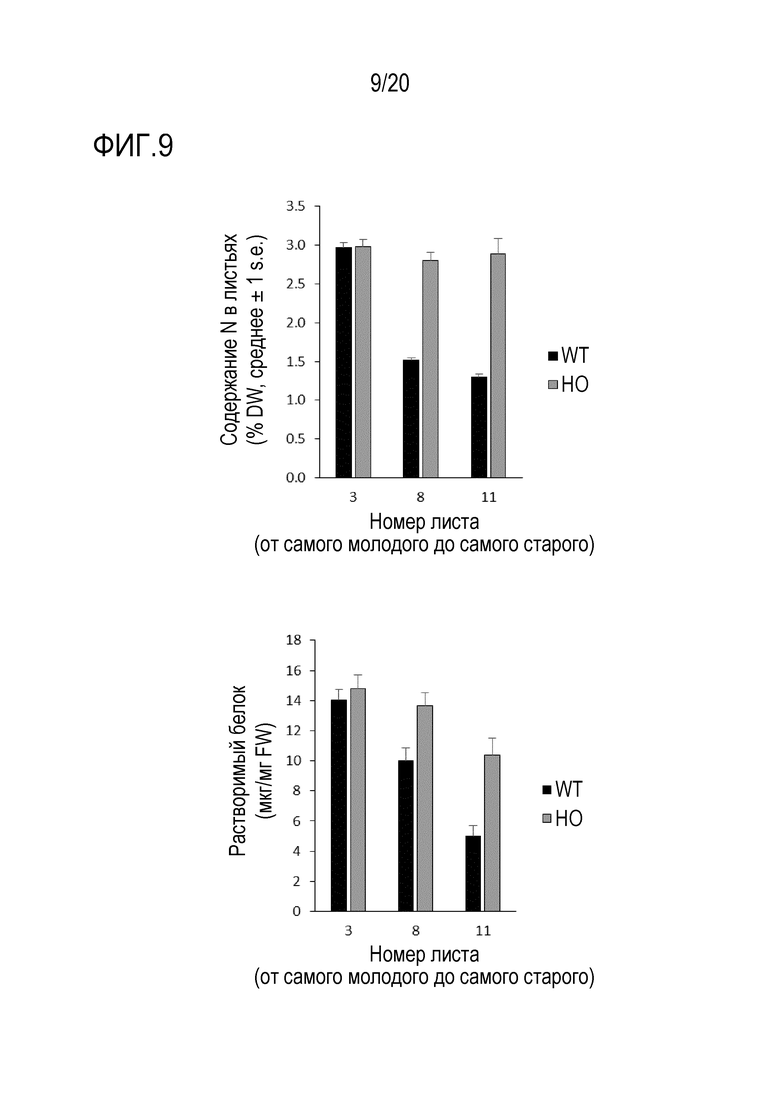
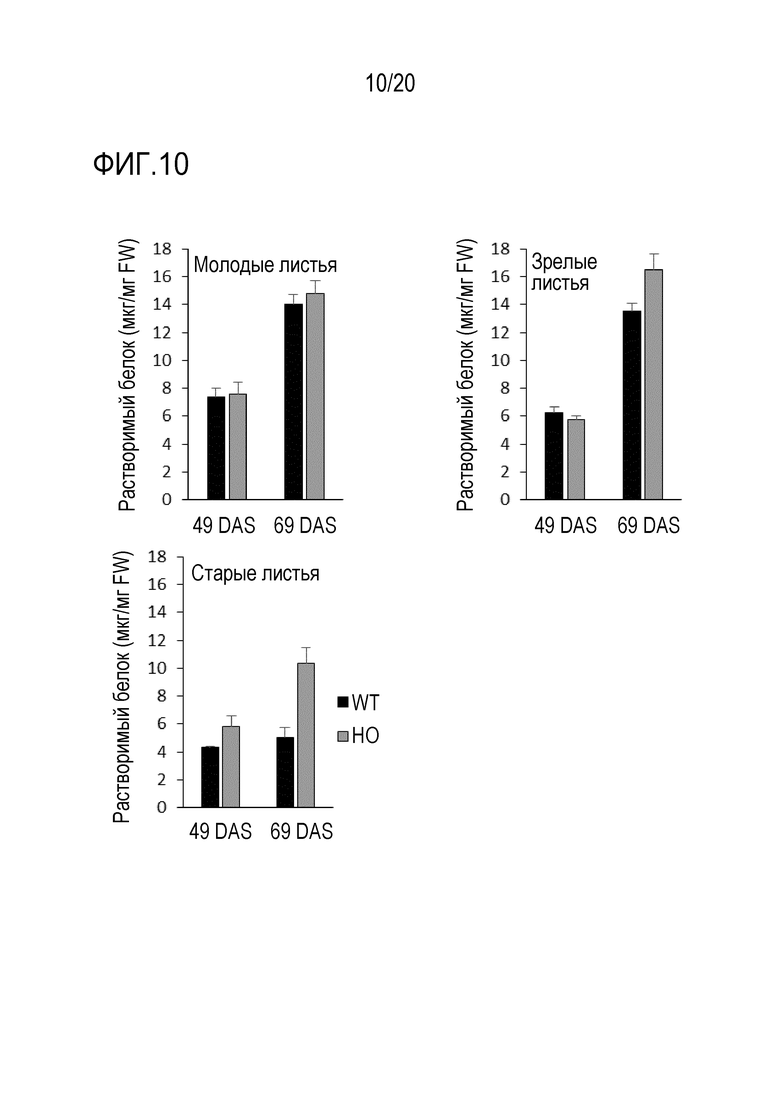
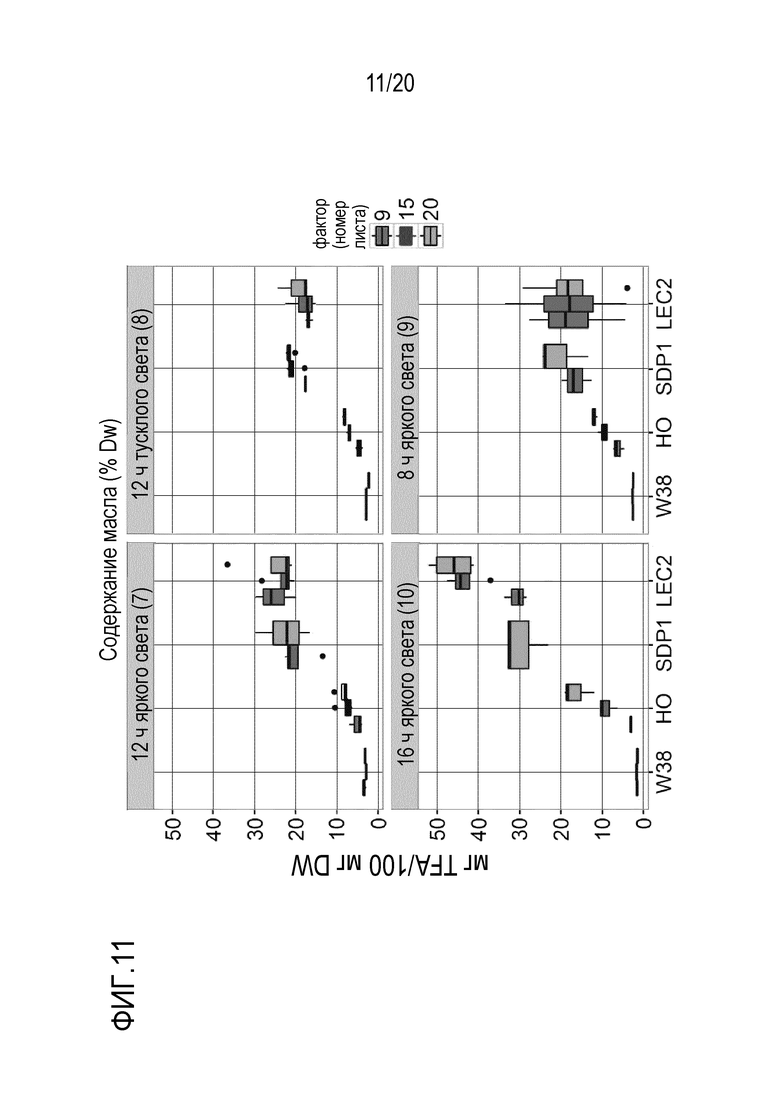
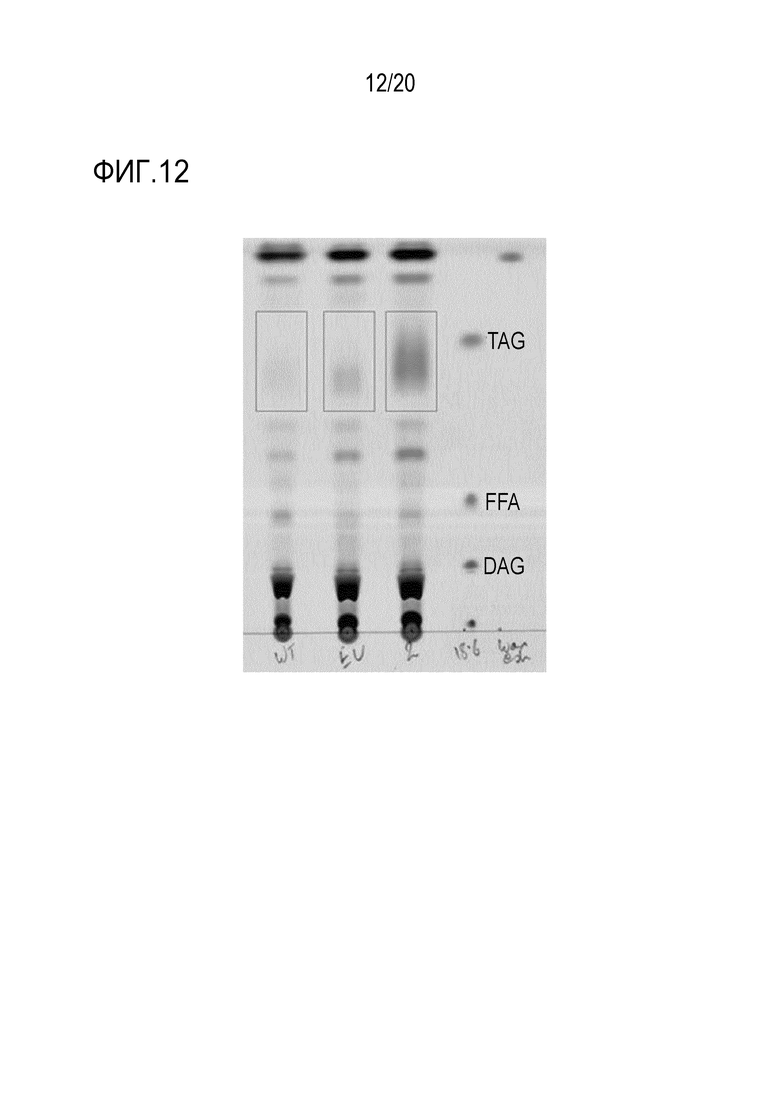
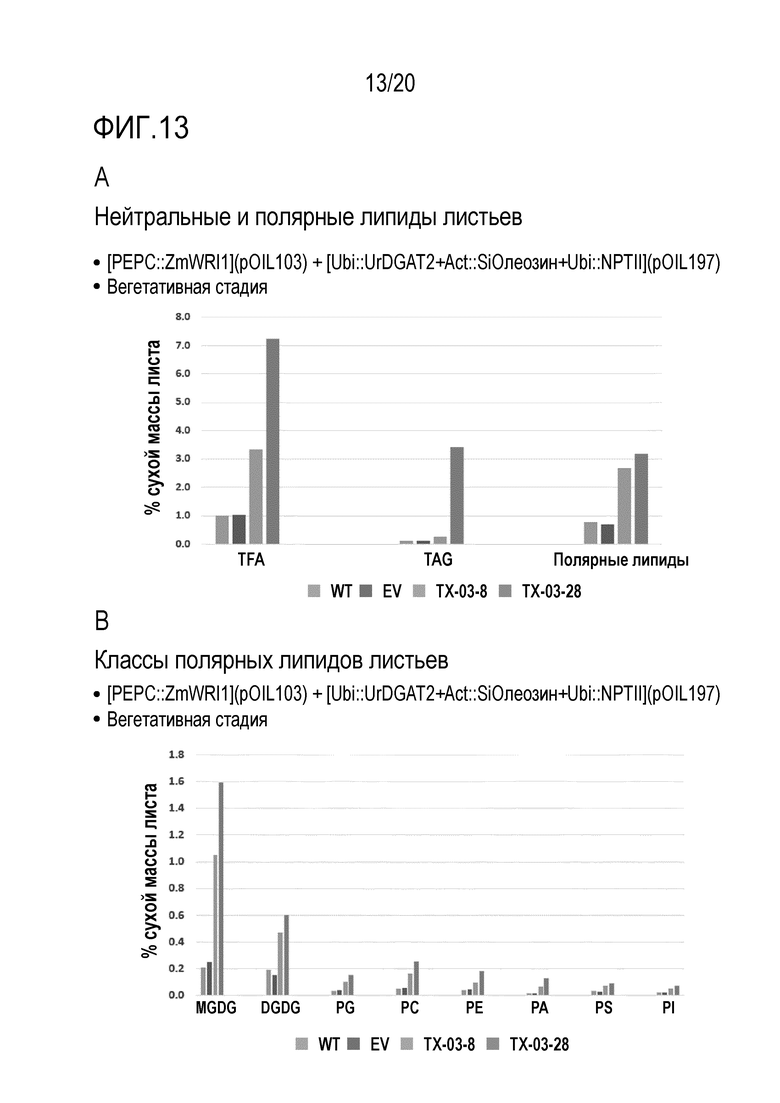
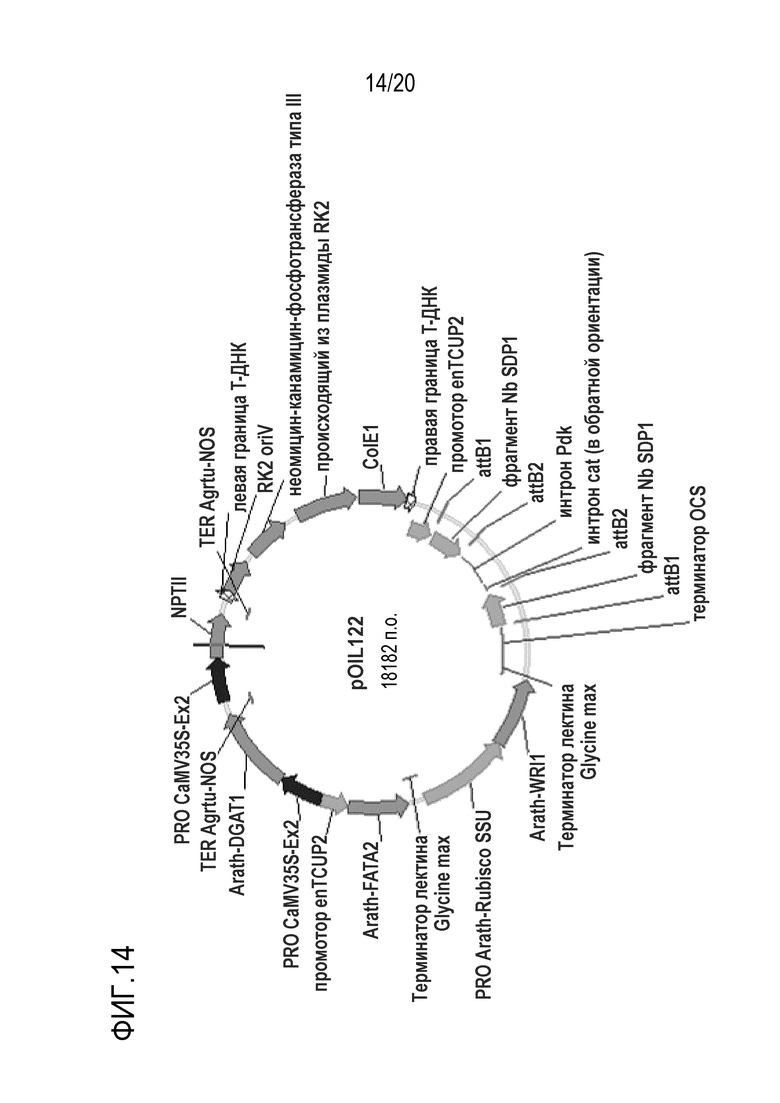
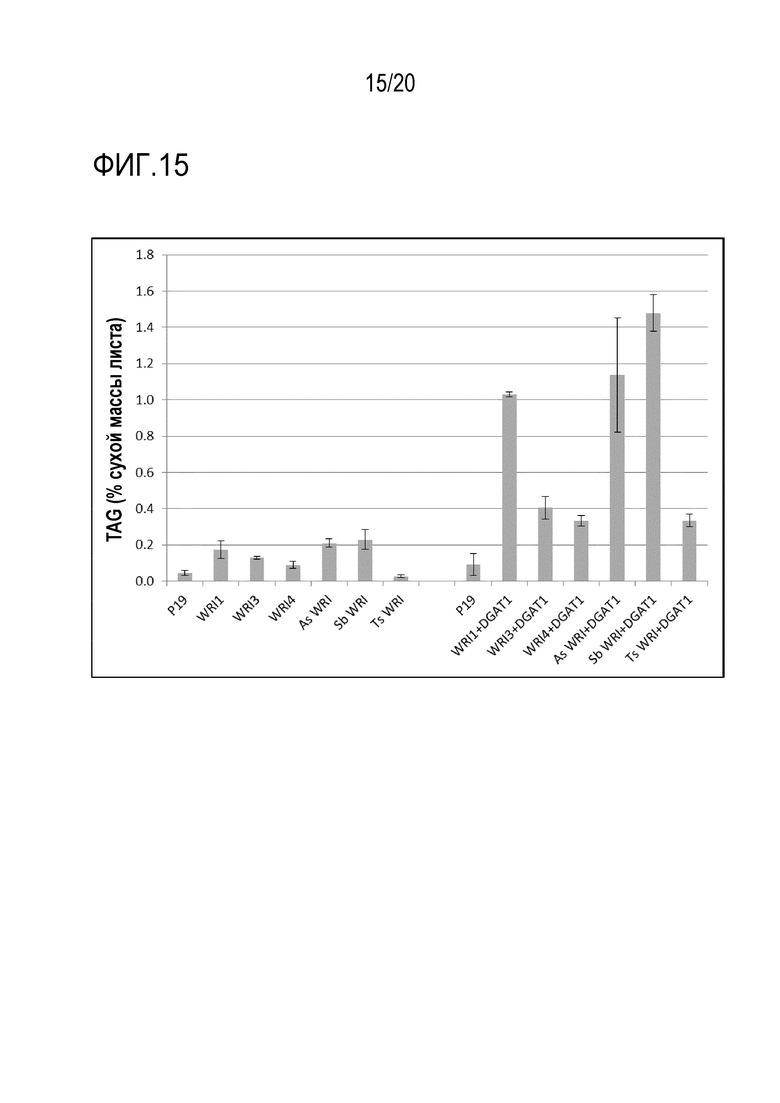
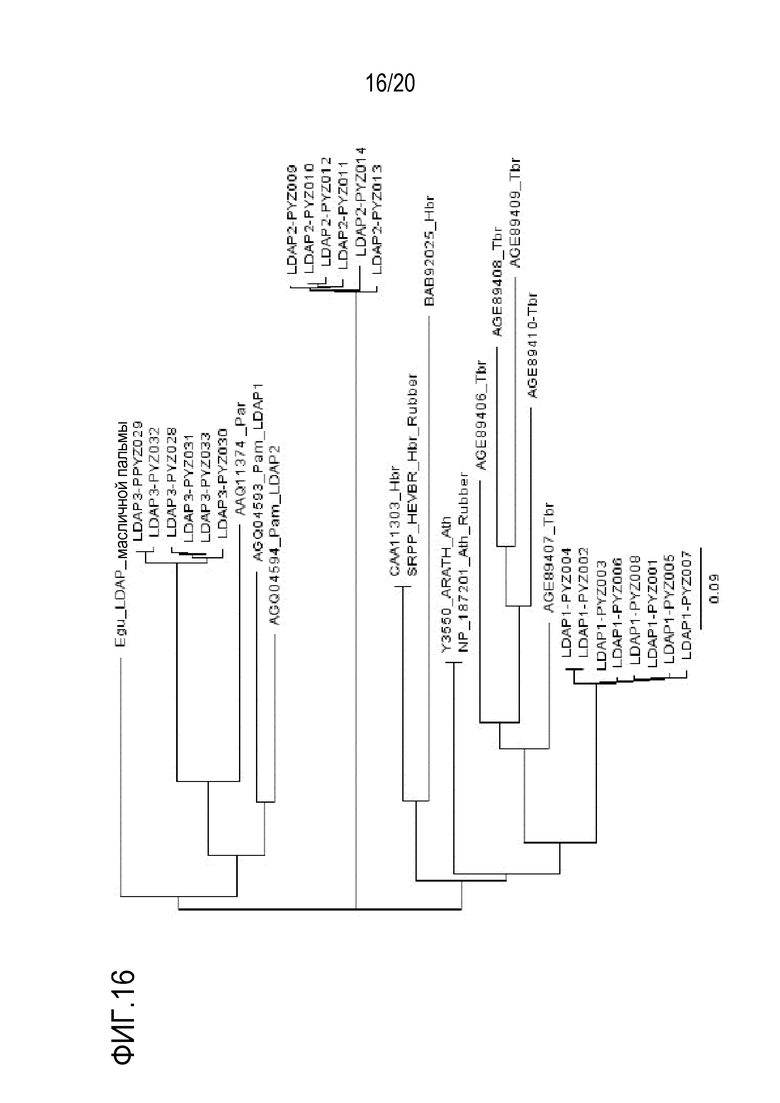
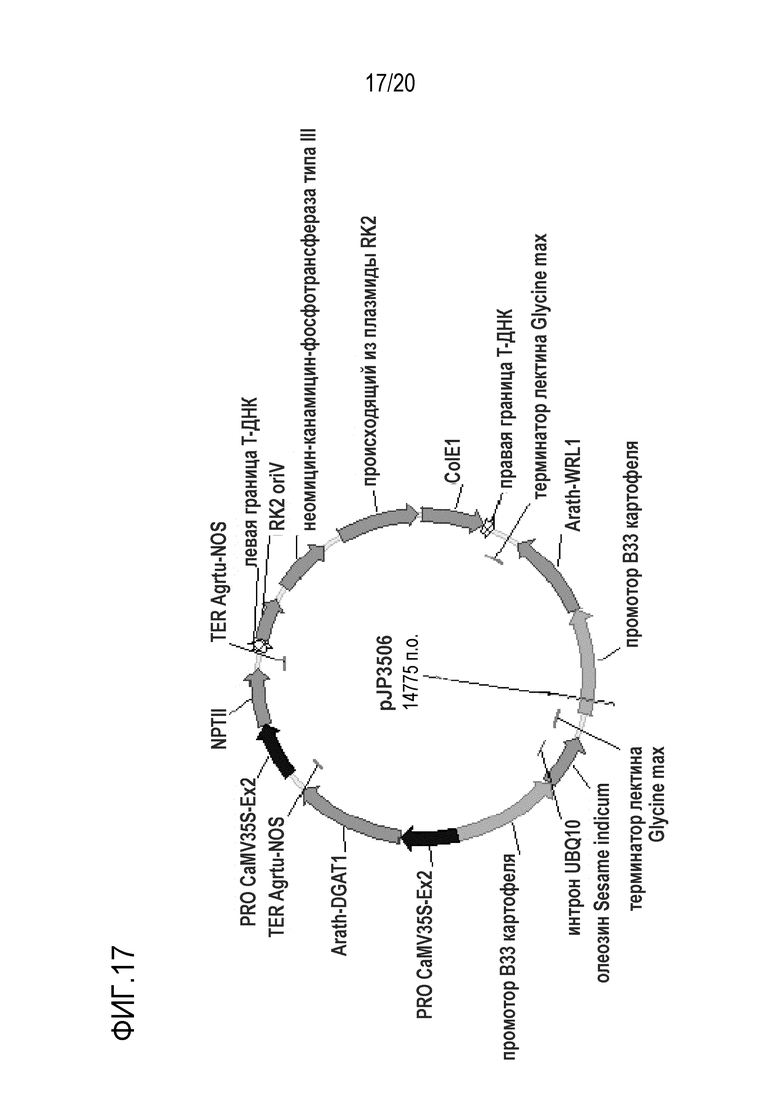
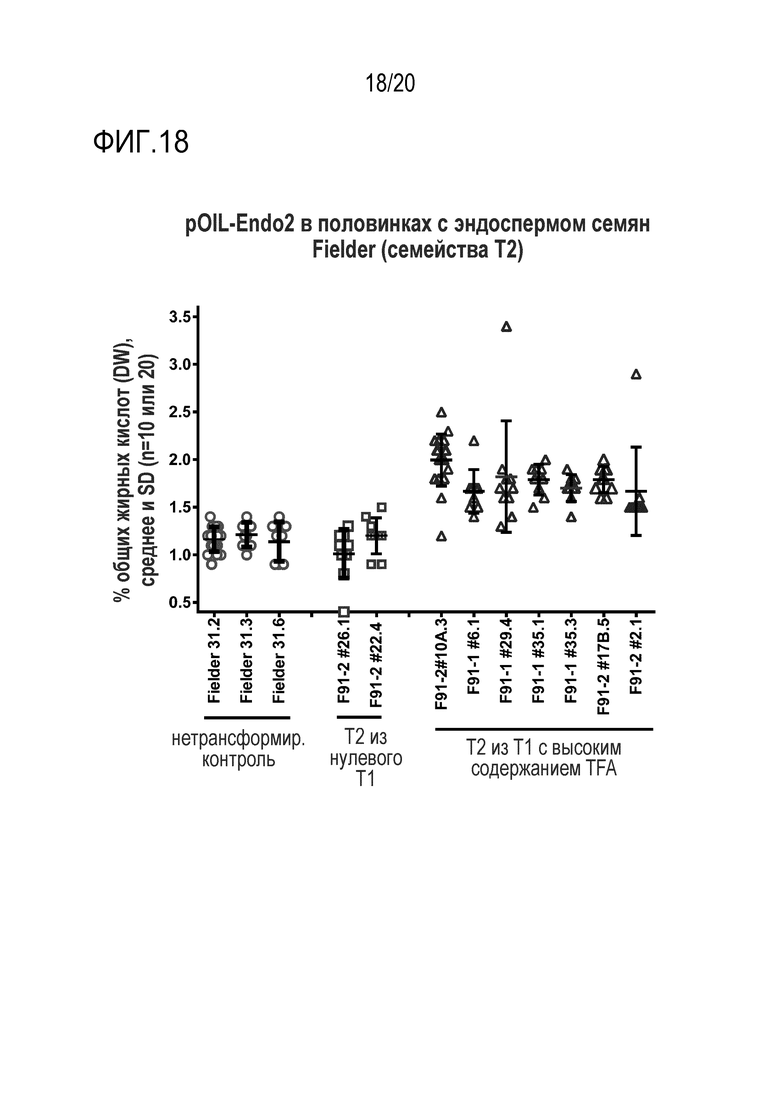


Изобретение относится к области биохимии, в частности к способу получения кормового продукта для животного. Также раскрыты способ кормления животного, кормовой продукт для животного, способ получения растения, способ производства промышленного продукта, способ получения экстрагированного липида, применение трансгенного растения вида Sorghum или Zea mays для производства промышленного продукта, способ получения топлива. Изобретение позволяет эффективно получать растения вида Sorghum, Zea mays с повышенным содержанием жирных кислот. 10 н. и 27 з.п. ф-лы, 20 ил., 31 табл., 18 пр.
1. Способ получения кормового продукта для животного, включающий стадию
(i) сбора вегетативных частей растений из растения вида Sorghum и/или Zea mays, имеющих (a) увеличенное содержание полипептида фактора транскрипции по сравнению с соответствующей вегетативной частью растения дикого типа, где полипептид фактора транскрипции представляет собой полипептид Wrinkled 1 (WRI1), экспрессируемый из экзогенного полинуклеотида, (b) увеличенное содержание полипептида ацилтрансферазы жирных кислот по сравнению с соответствующей вегетативной частью растения дикого типа, где ацилтрансфераза представляет собой диацилглицерин-ацилтрансферазу (DGAT), экспрессируемую из экзогенного полинуклеотида, и (c) общее содержание жирных кислот (TFA), включающее жирные кислоты, этерифицированные в форме триацилглицеринов (TAG), и жирные кислоты в форме липидов, отличных от TAG, где вегетативные части растений имеют содержание TFA по меньшей мере 5% (масс./масс. сухой массы) и имеют соотношение TAG/TFA (TTQ) между 0,01 и 0,60, или между 0,60 и 0,84, или между 0,84 и 0,95, и одну или несколько из стадий:
(ii) смешивания собранных частей растений по меньшей мере с одним другим кормовым ингредиентом,
(iii) упаковки в тюки собранных частей растений,
(iv) переработки собранных частей растений предпочтительно посредством рубки, нарезки, сушки, прессования или гранулирования частей растений в форму, пригодную для употребления животным и
(v) хранения собранных частей растений в условиях уменьшенного содержания кислорода в течение такого периода времени, что по меньшей мере некоторые из углеводов в частях растений подвергаются ферментации до органических кислот.
2. Способ по п.1, в котором применяют один или несколько или все из следующих признаков:
(i) вегетативные части растений собирают из растения между временем первого цветения растения и первого созревания семян,
(ii) растение вида Sorghum представляет собой растение Sorghum bicolor,
(iii) вегетативные части растений включают листья и стебли или их части,
(iv) вегетативные части растений имеют среднее содержание TFA приблизительно 6%, или приблизительно 8%, или приблизительно 9%, или приблизительно 10% (масс./масс. сухой массы),
(v) содержание TFA в вегетативных частях растений включает содержание олеиновой кислоты, которое увеличено по меньшей мере на 2% или по меньшей мере на 3% относительно содержания олеиновой кислоты в соответствующей вегетативной части растения дикого типа,
(vi) содержание TFA в вегетативных частях растений включает содержание пальмитиновой кислоты, которое увеличено по меньшей мере на 2% или по меньшей мере на 3% относительно содержания пальмитиновой кислоты в соответствующей вегетативной части растения дикого типа,
(vii) содержание TFA в вегетативных частях растений включает содержание α-линолеиновой кислоты (ALA), которое уменьшено по меньшей мере на 2% или по меньшей мере на 3% относительно содержания ALA в соответствующей вегетативной части растения дикого типа,
(viii) вегетативные части растений имеют увеличенное содержание растворимого белка по сравнению с соответствующей вегетативной частью растения дикого типа,
(ix) вегетативные части растений имеют увеличенное содержание азота по сравнению с соответствующей вегетативной частью растения дикого типа,
(x) вегетативные части растений имеют уменьшенное соотношение углерод:азот по сравнению с соответствующей вегетативной частью растения дикого типа,
(xi) листья растения вида Sorghum и/или Zea mays имеют увеличенную способность к фотосинтезу по сравнению с соответствующими листьями дикого типа,
(xii) вегетативные части растений имеют уменьшенное общее содержание пищевых волокон (TDF) по сравнению с соответствующей вегетативной частью растения дикого типа,
(xiii) вегетативные части растений имеют увеличенное содержание углерода по сравнению с соответствующей вегетативной частью растения дикого типа,
(xiv) вегетативные части растений дополнительно имеют увеличенное содержание полипептида фактора транскрипции по сравнению с соответствующей вегетативной частью растения дикого типа, где полипептид фактора транскрипции выбран из группы, состоящей из листовой семядоли 1 (LEC1), LEC1-подобного, листовой семядоли 2 (LEC2), BABY BOOM (BBM), FUS3, ABI3, ABI4, ABI5, Dof4 и Dof11, или из группы, состоящей из MYB73, bZIP53, AGL15, MYB115, MYB118, TANMEI, WUS, GFR2a1, GFR2a2 и PHR1,
(xv) вегетативные части растений дополнительно имеют увеличенное содержание полипептида ацилтрансферазы жирных кислот по сравнению с соответствующей вегетативной частью растения дикого типа, где ацилтрансфераза представляет собой фосфолипид:диацилглицерин-ацилтрансферазу (PDAT),
(xvi) вегетативные части растений имеют уменьшенное содержание полипептида TAG-липазы по сравнению с соответствующей вегетативной частью растения дикого типа,
(xvii) вегетативные части растений имеют уменьшенное содержание полипептида тригалактозилдиацилглицерина (TGD) по сравнению с соответствующей вегетативной частью растения дикого типа,
(xviii) вегетативные части растений имеют увеличенное содержание полипептида покрытия масляных телец (OBC) или ассоциированного с липидными капельками полипептида (LDAP) по сравнению с соответствующей вегетативной частью растения дикого типа,
(xix) вегетативные части растений имеют увеличенное общее содержание белка по сравнению с соответствующей вегетативной частью растения дикого типа,
(xx) вегетативные части растений имеют увеличенное содержание хлорофилла по сравнению с соответствующей вегетативной частью растения дикого типа,
(xxi) вегетативные части растений имеют увеличенное содержание энергии в пересчете на массу по сравнению с соответствующей вегетативной частью растения дикого типа,
(xxii) вегетативные части растений имеют увеличенное содержание фосфолипидов и/или галактолипидов, предпочтительно увеличенное содержание моногалактозил-диглицерида (MDGD) и/или увеличенное содержание дигалактозил-диглицерида (DGDG), по сравнению с соответствующей вегетативной частью растения дикого типа,
(xxiii) TTQ составляет приблизительно 0,1, или приблизительно 0,2, или приблизительно 0,3, или приблизительно 0,4, или приблизительно 0,5, или приблизительно 0,6, или приблизительно 0,65, или приблизительно 0,7, или приблизительно 0,75, или приблизительно 0,8, или приблизительно 0,81, или приблизительно 0,82, или приблизительно 0,83, или приблизительно 0,84, или приблизительно 0,85, или приблизительно 8,6, или приблизительно 8,7, или приблизительно 8,8, или приблизительно 8,9, или приблизительно 0,9, или приблизительно 0,91, или приблизительно 0,92, или приблизительно 0,93, или приблизительно 0,94, или приблизительно 0,95,
(xxiv) по меньшей мере один другой кормовой ингредиент содержит одно или несколько или все из: съедобных питательных макровеществ, витаминов, минералов (таких как кальций, фосфор, магний и сера), сена, такого как люцерновое сено, пивной дробины, жмыховой муки (из канолы или сои), хлопчатника, мелассы, дополнительных аминокислот (таких как лизин и метионин) и небелковых источников азота (таких как мочевина),
(xxv) период времени составляет между одной неделей и 52 неделями,
(xxvi) органические кислоты включают уксусную кислоту, пропионовую кислоту или масляную кислоту или любую их комбинацию,
(xxvii) кормовой продукт представляет собой силос, гранулы или сено и
(xxviii) вегетативные части растений хранят в течение некоторого периода времени перед смешиванием по меньшей мере с одним другим кормовым ингредиентом,
в каждом случае, когда соответствующую часть растения дикого типа собирают из растения вида Sorghum и/или Zea mays дикого типа на такой же стадии роста.
3. Способ по п.1 или 2, в котором применяют один или несколько или все из следующих признаков:
(i) вегетативные части растений представляют собой листья и/или стебли или их части, имеющие одно или несколько из увеличенного содержания углерода, увеличенного содержания энергии, увеличенного содержания растворимого белка, уменьшенного содержания крахмала, уменьшенного общего содержания пищевых волокон (TDF) и увеличенного содержания азота, каждое в пересчете на массу по сравнению с соответствующим листом или стеблем дикого типа или их частями, из растения вида Sorghum и/или Zea mays дикого типа на такой же стадии роста,
(ii) содержание TFA в вегетативных частях растений составляет по меньшей мере приблизительно 6%, по меньшей мере приблизительно 7%, по меньшей мере приблизительно 8%, по меньшей мере приблизительно 9%, по меньшей мере приблизительно 10%, по меньшей мере приблизительно 11%, по меньшей мере приблизительно 12%, по меньшей мере приблизительно 15%, по меньшей мере приблизительно 20%, по меньшей мере приблизительно 25%, по меньшей мере приблизительно 30%, по меньшей мере приблизительно 35%, по меньшей мере приблизительно 40%, по меньшей мере приблизительно 45%, по меньшей мере приблизительно 50%, по меньшей мере приблизительно 55%, по меньшей мере приблизительно 60%, по меньшей мере приблизительно 65%, по меньшей мере приблизительно 70%, между 8% и 75%, между 6% и 20%, между 10% и 75%, между 11% и 75%, между приблизительно 15% и 75%, между приблизительно 20% и 75%, между приблизительно 30% и 75%, между приблизительно 40% и 75%, между приблизительно 50% и 75%, между приблизительно 60% и 75% или между приблизительно 25% и 50% (масс./масс. сухой массы) TFA,
(iii) жирные кислоты, этерифицированные в форме TAG, в вегетативных частях растений составляют по меньшей мере приблизительно 1%, по меньшей мере приблизительно 2%, по меньшей мере приблизительно 3%, по меньшей мере приблизительно 4%, по меньшей мере приблизительно 5%, по меньшей мере приблизительно 6%, по меньшей мере приблизительно 7%, по меньшей мере приблизительно 8%, по меньшей мере приблизительно 9%, по меньшей мере приблизительно 10%, по меньшей мере приблизительно 11%, по меньшей мере приблизительно 12%, по меньшей мере приблизительно 15%, по меньшей мере приблизительно 20%, по меньшей мере приблизительно 25%, по меньшей мере приблизительно 30%, по меньшей мере приблизительно 35%, по меньшей мере приблизительно 40%, по меньшей мере приблизительно 45%, по меньшей мере приблизительно 50%, по меньшей мере приблизительно 55%, по меньшей мере приблизительно 60%, по меньшей мере приблизительно 65%, по меньшей мере приблизительно 70%, между 6% и 20%, между 8% и 75%, между 10% и 75%, между 11% и 75%, между приблизительно 15% и 75%, между приблизительно 20% и 75%, между приблизительно 30% и 75%, между приблизительно 40% и 75%, между приблизительно 50% и 75%, между приблизительно 60% и 75% или между приблизительно 25% и 50% (масс./масс. сухой массы),
(iv) вегетативные части растений имеют уменьшенное содержание полипептида SDP1 по сравнению с соответствующей вегетативной частью растения дикого типа,
(v) вегетативные части растений имеют увеличенное содержание полипептида LEC2 по сравнению с соответствующей вегетативной частью растения дикого типа,
(vi) вегетативные части растений имеют увеличенное содержание полипептида PDAT, уменьшенное содержание полипептида TGD и уменьшенное содержание полипептида SDP1, каждое по сравнению с соответствующей вегетативной частью растения дикого типа,
(vii) вегетативные части растений имеют уменьшенное содержание TAG-липазы, такой как TAG-липаза SDP1, уменьшенное содержание полипептида TGD, такого как полипептид TGD5, и, необязательно, уменьшенное содержание полипептида TST, такого как полипептид TST1, где каждое уменьшение присутствует по сравнению с соответствующей вегетативной частью растения дикого типа.
4. Способ кормления животного, включающий предоставление животному вегетативных частей растений из растения вида Sorghum и/или Zea mays, имеющих (a) увеличенное содержание полипептида фактора транскрипции по сравнению с соответствующей вегетативной частью растения дикого типа, где полипептид фактора транскрипции представляет собой полипептид Wrinkled 1 (WRI1), экспрессируемый из экзогенного полинуклеотида, (b) увеличенное содержание полипептида ацилтрансферазы жирных кислот по сравнению с соответствующей вегетативной частью растения дикого типа, где ацилтрансфераза представляет собой диацилглицерин-ацилтрансферазу (DGAT), экспрессируемую из экзогенного полинуклеотида, и (c) общее содержание жирных кислот (TFA), включающее жирные кислоты, этерифицированные в форме триацилглицеринов (TAG), и жирные кислоты в форме липидов, отличных от TAG, где вегетативные части растений имеют содержание TFA по меньшей мере 5% (масс./масс. сухой массы) и имеют соотношение TAG/TFA (TTQ) между 0,01 и 0,60, или между 0,60 и 0,84, или между 0,84 и 0,95.
5. Способ по п.4, в котором применяют один или несколько из следующих признаков:
(i) вегетативные части растений содержатся в растении вида Sorghum и/или Zea mays, растущем в поле,
(ii) вегетативные части растений собирают из растения вида Sorghum и/или Zea mays и/или смешивают по меньшей мере с одним другим кормовым ингредиентом,
(iii) вегетативные части растений перерабатывали после сбора предпочтительно посредством рубки, нарезки, сушки, прессования или гранулирования частей растений в форму, пригодную для употребления животным,
(iv) собранные части растений хранили в условиях уменьшенного содержания кислорода в течение такого периода времени, что по меньшей мере некоторые из углеводов в частях растений подвергаются ферментации до органических кислот до предоставления животному, и
(v) собранные части растений хранят в течение некоторого периода времени между их сбором и предоставлением животному.
6. Способ по п.4 или 5, где животное употребляет внутрь увеличенное количество азота, белка, углерода и/или энергетического потенциала или уменьшенное количество крахмала или TDF относительно того, когда животное употребляет внутрь такое же количество в пересчете на сухую массу соответствующего кормового продукта, полученного с использованием эквивалентного количества растения дикого типа вида Sorghum и/или Zea mays или его частей.
7. Способ по любому из пп. 4-6, в котором применяют один или несколько или все из признаков по п.2 или 3.
8. Способ по п. 1 или 4, в котором вегетативные части растений имеют содержание TFA между 6% и 20% (масс./масс. сухой массы).
9. Кормовой продукт для животного, содержащий собранные вегетативные части растений из растения вида Sorghum и/или Zea mays, имеющие (a) увеличенное содержание полипептида фактора транскрипции по сравнению с соответствующей вегетативной частью растения дикого типа, где полипептид фактора транскрипции представляет собой полипептид Wrinkled 1 (WRI1), экспрессируемый из экзогенного полинуклеотида, (b) увеличенное содержание полипептида ацилтрансферазы жирных кислот по сравнению с соответствующей вегетативной частью растения дикого типа, где ацилтрансфераза представляет собой диацилглицерин-ацилтрансферазу (DGAT), экспрессируемую из экзогенного полинуклеотида, и (c) общее содержание жирных кислот (TFA), включающее жирные кислоты, этерифицированные в форме триацилглицеринов (TAG), и жирные кислоты в форме липидов, отличных от TAG, где вегетативные части растений имеют содержание TFA по меньшей мере 5% (масс./масс. сухой массы) и имеют соотношение TAG/TFA (TTQ) между 0,01 и 0,60, или между 0,60 и 0,84, или между 0,84 и 0,95, где применяют один или несколько из следующих признаков:
(i) собранные части растений смешаны по меньшей мере с одним другим кормовым ингредиентом,
(ii) собранные части растений упакованы в тюки после сбора,
(iii) собранные части растений переработаны предпочтительно посредством рубки, нарезки, сушки, прессования или гранулирования частей растений в форму, пригодную для употребления животным и
(iv) собранные части растений хранили в условиях уменьшенного содержания кислорода в течение такого периода времени, что по меньшей мере некоторые из углеводов в частях растений подверглись ферментации до органических кислот.
10 Кормовой продукт по п.9, который представляет собой силос, гранулы или сено.
11. Кормовой продукт по п.9 или 10, в котором применяют один или несколько или все из признаков по п.2 или 3.
12. Кормовой продукт по любому из пп. 9-11, в котором вегетативные части растений имеют содержание TFA между 6% и 20% (масс./масс. сухой массы).
13. Способ получения растения вида Sorghum или Zea mays, в геном которого интегрирован набор экзогенных полинуклеотидов, включающий стадии
i) скрещивания двух материнских растений, где одно растение содержит по меньшей мере один из экзогенных полинуклеотидов, выбранных из (а) фактора транскрипции Wrinkled 1 (WRI1) и (b) диацилглицерин-ацилтрансферазы (DGAT), и другое растение содержит по меньшей мере один из экзогенных полинуклеотидов, выбранных из (а) фактора транскрипции Wrinkled 1 (WRI1) и (b) диацилглицерин-ацилтрансферазы (DGAT), и при этом два материнских растения содержат между собой два экзогенных полинуклеотида,
ii) скрининга одного или нескольких растений-потомков скрещивания на предмет присутствия или отсутствия двух экзогенных полинуклеотидов и
iii) отбора растения-потомка, содержащего два экзогенных полинуклеотида,
с получением таким образом растения.
14. Способ получения нефтепродукта, включающий стадии
(i) обработки в реакторе композиции, содержащей:
(a) вегетативные части растений вида Sorghum или Zea mays, имеющие (a) увеличенное содержание полипептида фактора транскрипции по сравнению с соответствующей вегетативной частью растения дикого типа, где полипептид фактора транскрипции представляет собой полипептид Wrinkled 1 (WRI1), экспрессируемый из экзогенного полинуклеотида, (b) увеличенное содержание полипептида ацилтрансферазы жирных кислот по сравнению с соответствующей вегетативной частью растения дикого типа, где ацилтрансфераза представляет собой диацилглицерин-ацилтрансферазу (DGAT), экспрессируемую из экзогенного полинуклеотида, и (c) общее содержание жирных кислот (TFA) в которых включает жирные кислоты, этерифицированные в форме триацилглицеринов (TAG), и жирные кислоты в форме липидов, отличных от TAG, где вегетативная часть растения имеет содержание TFA по меньшей мере 5% (масс./масс. сухой массы) и имеет соотношение TAG/TFA (TTQ) между 0,01 и 0,60, или между 0,60 и 0,84, или между 0,84 и 0,95,
(b) растворитель, который содержит воду, спирт или и то, и другое, и
(c) необязательно, катализатор,
где обработка включает нагревание композиции при температуре между приблизительно 50°C и приблизительно 450°C и давлении от 5 до 350 бар на протяжении периода от 1 до 120 минут в окислительной, восстановительной или инертной среде,
(ii) выделения нефтепродукта из реактора с выходом по меньшей мере 35 мас.% в пересчете на сухую массу вегетативных частей растений,
с получением таким образом нефтепродукта.
15. Способ по п.14, характеризующийся одним или нескольким или всем из:
(i) вегетативные части растений имеют сухую массу по меньшей мере 1 кг,
(ii) вегетативные части растений имеют содержание TFA и/или общее содержание неполярных липидов по меньшей мере 10%, по меньшей мере 15%, по меньшей мере 20%, приблизительно 25%, приблизительно 30%, приблизительно 35% или между 30% и 75% в пересчете на сухую массу,
(iii) концентрация твердых веществ в композиции составляет между 5% и 90%
(iv) катализатор содержит NaOH или KOH или оба предпочтительно в концентрации 0,1-2 М,
(v) время обработки составляет между 1 и 60 минут, предпочтительно между 10 и 60 минут, предпочтительно между 15 и 30 минут,
(vi) если растворитель представляет собой воду, способ обеспечивает выход нефтепродукта между минимум 36%, 37%, 38%, 39% или 40% и максимум 55% или 60% по массе в пересчете на сухую массу в вегетативных частях растений,
(vii) если растворитель содержит спирт, способ обеспечивает выход нефтепродукта между минимум 36%, 37%, 38%, 39% или 40% и максимум 65% или 70% по массе в пересчете на сухую массу вегетативных частей растений,
(viii) если растворитель содержит приблизительно 80% воды, нефтепродукт содержит приблизительно 30% C13-C22 углеводородных соединений,
(ix) если растворитель содержит приблизительно 50% метанола, нефтепродукт содержит приблизительно 50% метиловых эфиров жирных кислот (FAME),
(x) содержание воды в выделенном нефтепродукте составляет менее чем приблизительно 15% по массе,
(xi) выход нефтепродукта по меньшей мере на 2 мас.% больше, чем выход соответствующего способа с использованием соответствующих вегетативных частей растений, в которых содержание неполярных липидов составляет менее чем 2% в пересчете на сухую массу, и
(xii) вегетативные части растений на стадии (i)(a) физически обрабатывают посредством одного или нескольких из сушки, рубки, шинковки, помола, вальцевания, прессования, дробления или перетирания.
16. Способ по п.14 или 15, дополнительно включающий одно или несколько из:
(i) гидродезоксигенации выделенного нефтепродукта,
(ii) обработки выделенного нефтепродукта водородом с целью уменьшения уровней кетонов или сахаров в нефтепродукте,
(iii) получения синтетического газа из выделенного нефтепродукта и
(iv) фракционирования выделенного нефтепродукта с целью получения одного или нескольких из мазута, дизельного топлива, керосина или бензина.
17. Способ по любому из пп. 14-16, где вегетативные части растений включают листья, стебли растения или и то, и другое.
18. Способ по п. 14, в котором вегетативная часть растения имеет содержание TFA между 6% и 20% (масс./масс. сухой массы).
19. Способ производства промышленного продукта, включающий стадии:
i) получения трансгенного растения вида Sorghum или Zea mays, части растения или его семени, включающего (a) увеличенное содержание полипептида фактора транскрипции по сравнению с соответствующей вегетативной частью растения дикого типа, где полипептид фактора транскрипции представляет собой полипептид Wrinkled 1 (WRI1), экспрессируемый из экзогенного полинуклеотида, (b) увеличенное содержание полипептида ацилтрансферазы жирных кислот по сравнению с соответствующей вегетативной частью растения дикого типа, где ацилтрансфераза представляет собой диацилглицерин-ацилтрансферазу (DGAT), экспрессируемую из экзогенного полинуклеотида, и (c) вегетативную часть растения, общее содержание жирных кислот (TFA) в которой включает жирные кислоты, этерифицированные в форме триацилглицеринов (TAG), и жирные кислоты в форме липидов, отличных от TAG, где вегетативная часть растения имеет содержание TFA по меньшей мере 5% (масс./масс. сухой массы) и имеет соотношение TAG/TFA (TTQ) между 0,01 и 0,60, или между 0,60 и 0,84, или между 0,84 и 0,95, и
ii) либо:
a) перевода по меньшей мере части липида в клетке, растении или его части или семени со стадии i) в промышленный продукт с применением нагревания, химических или ферментных средств или любой их комбинации к липиду in situ в растении или его части или семени или
b) физической обработки растения или его части или семени со стадии i) и одновременного или последующего перевода по меньшей мере части липида в обрабатываемой растении или его части или семени в промышленный продукт с применением нагревания, химических или ферментных средств или любой их комбинации к липиду в обрабатываемой растении или его части или семени, и
iii) выделения промышленного продукта
с получением таким образом промышленного продукта, где промышленный продукт представляет собой углеводородный продукт, содержащий сложные эфиры жирных кислот, предпочтительно метиловые эфиры жирных кислот и/или этиловые эфиры жирных кислот, алкан, такой как метан, этан или алкан с более длинной цепью, смесь алканов с более длинной цепью, алкен, биотопливо, монооксид углерода и/или газообразный водород, биоспирт, такой как этанол, пропанол или бутанол, биоуголь или комбинация монооксида углерода, водорода и биоугля.
20. Способ по п.19, где часть растения представляет собой вегетативную часть растения.
21. Способ по п.19 или 20, дополнительно включающий стадии:
(a) экстракции по меньшей мере части неполярного липида, содержащегося в растении или его части или семени в форме неполярного липида, и
(b) выделения экстрагированного неполярного липида,
где стадии (a) и (b) выполняют до стадии перевода по меньшей мере части липида в растении или его части или семени в промышленный продукт.
22. Способ получения экстрагированного липида, включающий стадии:
i) получения трансгенного растения вида Sorghum или Zea mays, части растения или его семени, включающего (a) увеличенное содержание полипептида фактора транскрипции по сравнению с соответствующей вегетативной частью растения дикого типа, где полипептид фактора транскрипции представляет собой полипептид Wrinkled 1 (WRI1), экспрессируемый из экзогенного полинуклеотида, (b) увеличенное содержание полипептида ацилтрансферазы жирных кислот по сравнению с соответствующей вегетативной частью растения дикого типа, где ацилтрансфераза представляет собой диацилглицерин-ацилтрансферазу (DGAT), экспрессируемую из экзогенного полинуклеотида, и (c) вегетативную часть растения, общее содержание жирных кислот (TFA) в которой включает жирные кислоты, этерифицированные в форме триацилглицеринов (TAG), и жирные кислоты в форме липидов, отличных от TAG, где вегетативная часть растения имеет содержание TFA по меньшей мере 5% (масс./масс. сухой массы) и имеет соотношение TAG/TFA (TTQ) между 0,01 и 0,60, или между 0,60 и 0,84, или между 0,84 и 0,95,
ii) экстракции липида из клетки, растения или его части или семени и
iii) выделения экстрагированного липида,
с получением таким образом экстрагированного липида.
23. Способ по п.22, включающий экстракцию липида посредством одного или нескольких из сушки, вальцовки, прессования, дробления или перетирания растения или его части или семени и/или очистки экстрагированного липида или масла из семян.
24. Способ по п.23, включающий применение органического растворителя в процессе экстракции для экстракции масла.
25. Способ по любому из пп. 22-24, включающий выделение экстрагированного липида посредством сбора его в емкость и/или одного или нескольких из дегуммации, дезодорирования, обесцвечивания, сушки, фракционирования экстрагированного липида, удаления по меньшей мере некоторых восков и/или сложных эфиров восков из экстрагированного липида или анализа состава жирных кислот экстрагированного липида.
26. Способ по любому из пп. 22-24, где объем экстрагированного липида или масла составляет по меньшей мере 1 литр.
27. Способ по любому из пп. 22-24, где применяют один или несколько или все из следующих признаков:
(i) экстрагированный липид или масло содержит триацилглицерины, где триацилглицерины составляют по меньшей мере 90%, предпочтительно по меньшей мере 95% или по меньшей мере 96% экстрагированного липида или масла,
(ii) экстрагированный липид или масло содержит свободные стеролы, стероильные сложные эфиры, стероилгликозиды, воски или сложные эфиры восков или любую их комбинацию и
(iii) общее содержание и/или состав стеролов в экстрагированном липиде или масле значительно отличается от содержания и/или состава стеролов в экстрагированном липиде или масле, полученном из соответствующего растения или его части или семени.
28. Способ по любому из пп. 22-27, дополнительно включающий перевод экстрагированного липида в промышленный продукт.
29. Способ по любому из пп. 19-28, где часть растения представляет собой воздушную часть растения или зеленую часть растения, предпочтительно вегетативную часть растения, такую как лист или стебель растения.
30. Способ по любому из пп. 19-29, где стадия получения растения или его части включает стадию сбора урожая растения или его части с помощью механического сборщика.
31. Способ по любому из пп. 19-30, где уровень липида в растении или его части или семени и/или в экстрагированном липиде или масле поддается определению посредством анализа с использованием газовой хроматографии метиловых сложных эфиров жирных кислот, полученных из экстрагированного липида или масла.
32. Способ по любому из пп. 19-31, где часть растения представляет собой вегетативную часть растения, имеющую общее содержание TAG по меньшей мере приблизительно 11%, по меньшей мере приблизительно 12%, по меньшей мере приблизительно 15%, по меньшей мере приблизительно 20%, по меньшей мере приблизительно 25%, по меньшей мере приблизительно 30%, по меньшей мере приблизительно 35%, по меньшей мере приблизительно 40%, по меньшей мере приблизительно 45%, по меньшей мере приблизительно 50%, по меньшей мере приблизительно 55%, по меньшей мере приблизительно 60%, по меньшей мере приблизительно 65%, по меньшей мере приблизительно 70%, между 6% и 20%, между 8% и 75%, между 10% и 75%, между 11% и 75%, между приблизительно 15% и 75%, между приблизительно 20% и 75%, между приблизительно 30% и 75%, между приблизительно 40% и 75%, между приблизительно 50% и 75%, между приблизительно 60% и 75% или между приблизительно 25% и 50% (масс./масс. сухой массы).
33. Применение трансгенного растения вида Sorghum или Zea mays, части растения или его семени, включающего (a) увеличенное содержание полипептида фактора транскрипции по сравнению с соответствующей вегетативной частью растения дикого типа, где полипептид фактора транскрипции представляет собой полипептид Wrinkled 1 (WRI1), экспрессируемый из экзогенного полинуклеотида, (b) увеличенное содержание полипептида ацилтрансферазы жирных кислот по сравнению с соответствующей вегетативной частью растения дикого типа, где ацилтрансфераза представляет собой диацилглицерин-ацилтрансферазу (DGAT), экспрессируемую из экзогенного полинуклеотида, и (c) вегетативную часть растения, общее содержание жирных кислот (TFA) в которой включает жирные кислоты, этерифицированные в форме триацилглицеринов (TAG), и жирные кислоты в форме липидов, отличных от TAG, где вегетативная часть растения имеет содержание TFA по меньшей мере 5% (масс./масс. сухой массы) и имеет соотношение TAG/TFA (TTQ) между 0,01 и 0,60, или между 0,60 и 0,84, или между 0,84 и 0,95, для производства промышленного продукта, где промышленный продукт является углеводородным продуктом, включающим сложные эфиры жирных кислот, предпочтительно метиловые сложные эфиры жирных кислот и/или этиловые сложные эфиры жирных кислот, алкан, такой как метан, этан или длинноцепочечный алкан, смесь алканов с более длинной цепью, алкен, биотопливо, газообразный монооксид углерода и/или водород, биоспирт, такой как этанол, пропанол, или бутанол, биоуголь или комбинация монооксида углерода, водорода и биоугля.
34. Способ получения топлива, включающий:
i) экстракцию липида в соответствии со способом по п. 21; и
ii) проведение реакции липида из стадии ii) со спиртом.
35. Способ по п. 34, включающий:
iii) смешивание алкиловых сложных эфиров с топливом на основе нефти.
36. Способ получения синтетического дизельного топлива, включающий:
i) экстракцию липида в соответствии со способом по п. 21; и
ii) перевод липида из стадии i) в бионефть посредством способа, включающего пиролиз или гидротермическую обработку, или в синтетический газ посредством газификации; и
iii) перевод бионефти в синтетическое дизельное топливо посредством способа, включающего фракционирование, предпочтительно, с отбором углеводородных соединений, которые конденсируются при температуре между приблизительно 150°C и приблизительно 200°C или между приблизительно 200°C и приблизительно 300oC, или перевод синтетического газа в биотопливо с использованием металлического катализатора или микробного катализатора.
37. Способ по п.36, где часть представляет собой вегетативную часть растения.
| WO 2016004473 A1, 14.01.2016 | |||
| US 20150337017 A1, 26.11.2015 | |||
| СМЕСИ ЖИРНЫХ КИСЛОТ И ИХ ПРИМЕНЕНИЕ | 2007 |
|
RU2483057C2 |
Авторы
Даты
2023-12-06—Публикация
2017-09-01—Подача