ОБЛАСТЬ ТЕХНИКИ, К КОТОРОЙ ОТНОСИТСЯ ИЗОБРЕТЕНИЕ
Настоящее изобретение относится к способам и механизмам продуцирования полипептидов нитрогеназы в митохондриях растительных клеток.
ПРЕДПОСЫЛКИ СОЗДАНИЯ ИЗОБРЕТЕНИЯ
Диазотрофные бактерии продуцируют аммиак из газообразного N2 посредством биологической фиксации азота (BNF), катализируемой ферментным комплексом, нитрогеназой. Однако потребности γr современного сельского хозяйства опережают этот источник фиксированного азота и поэтому промышленно производимые азотные удобрения находят широкое применение в сельском хозяйстве (Smil, 2002). Однако производство удобрений и их применение являются причинами загрязнений (Good и Beatty, 2011) наносящими вред экологии (Rockstrom et al., 2009). Большая часть удобрений, применяемых во всем мире, не усваивается культурами (Cui et al., 2013; de Bruijn, 2015), что приводит к поверхностному стоку удобрений, разрастанию сорняков и эвтрофикации водоемов (Good и Beatty, 2011). Возникающее в результате цветение воды, вызванное массовым развитием водорослей, снижает уровень кислорода, приводит к локальному экологическому ущербу и ущербу для расположенных в прибрежных водах коралловых рифов (De'ath et al., 2012; Glibert et al., 2014; Sutton et al., 2008). Кроме того, хотя избыточное использование удобрений и является проблемой во многих развитых странах, однако в некоторых регионах их доступность ограничивает урожайность (Mueller et al., 2012). Само производство удобрений требует значительных затрат энергии и около 100 миллиардов долларов в год.
Очевидно, что необходима разработка стратегий по снижению зависимости от азота, производимого промышленным способом. С этой целью понятие инженерия растений, способных осуществлять биологическую фиксацию азота, давно привлекает значительный интерес (Merrick и Dixon, 1984) и является главным предметом внимания недавних периодических изданий (de Bruijn, 2015; Oldroyd и Dixon, 2014). Возможные подходы включают: i) расширение симбиотических отношений диазотрофов от бобовых до злаковых (Santi et al., 2013), ii) реинженерию эндосимбиотических микроорганизмов, способных осуществлять фиксацию азота (Geddes et al., 2015), и iii) генную инженерию нитрогеназы в растительных клетках (Curatti и Rubio, 2014). Все эти подходы амбициозны и спекулятивны в результате технической сложности.
Нитрогеназа представляет собой ферментный комплекс, способный осуществлять биологическую фиксацию азота у диазотрофных бактерий, и требует мультигенного пути сборки для своего биосинтеза и функционирования, что подробно рассматривается в работах (Hu и Ribbe, 2013; Rubio и Ludden, 2008; Seefeldt et al., 2009). Компоненты канонической железо-молибденовой нитрогеназы включают каталитические белки, обозначенные NifD и NifK, и донор электронов NifH. В сборке нитрогеназы у диазотрофных бактерий участвуют около 12 других белков, в том числе в созревании, поддержке и встраивании кофакторов комплекса, в частности NifM, NifS, NifU, NifE, NifN, NifX, NifV, NifJ, NifY, NifF, NifZ и NifQ. Генетические повреждения, анализ комплементации между диазотрофными и недиазотрофными прокариотами и филогенетические анализы (Dos Santos et al., 2012; Temme et al., 2012; Wang et al., 2013) привели к подмножеству белков Nif (NifD, NifK, NifB, NifE и NifN), рассматриваемому как центральные компоненты, а другие считаются необходимыми для оптимизированной активности и считаются вспомогательными. Для сборки и функционирования нитрогеназы также требуются определенные биохимические условия. Прежде всего, нитрогеназа чрезвычайно чувствительна к кислороду (Robson и Postgate, 1980). Кроме того, для биосинтеза и функционирования каталитического центра металлопротеинов требуются большие количества ATP, восстановителя, легко доступных Fe, Mo, S-аденозилметионина и гомоцитрата (Hu и Ribbe, 2013; Rubio и Ludden, 2008). Все эти факторы усугубляют техническую сложность продуцирования функционального комплекса нитрогеназы в растительных клетках.
КРАТКОЕ ИЗЛОЖЕНИЕ СУЩЕСТВА ИЗОБРЕТЕНИЯ
Авторы настоящего изобретения обнаружили важность экспрессии NifD, которая устойчива к вторичному расщеплению/деградации в растительных клетках, принимая во внимание наблюдаемую трудность в продуцировании функционального NifD в растительных клетках.
Таким образом, в одном аспекте настоящее изобретение относится к растительной клетке, содержащей экзогенный полинуклеотид, который кодирует полипептид NifD (ND), который устойчив к расщеплению протеазой на сайте в аминокислотной последовательности, соответствующей аминокислотам 97-100 из SEQ ID NO:18.
Во взаимосвязанном аспекте настоящее изобретение относится к растительной клетке, содержащей экзогенный полинуклеотид, который кодирует полипептид NifD (ND), который содержит аминокислотную последовательность, отличную от RRNY (SEQ ID NO:101) в позициях, соответствующих аминокислотам 97-100 из SEQ ID NO:18.
В предпочтительном варианте осуществления, ND более устойчив к расщеплению протеазой на сайте в аминокислотной последовательности, соответствующей аминокислотам 97-100 из SEQ ID NO:18, чем соответствующий ND, который имеет аминокислотную последовательность RRNY (SEQ ID NO:101) в позициях, соответствующих аминокислотам 97-100 из SEQ ID NO:18.
В варианте осуществления вышеупомянутых аспектов, ND содержит митохондриальный нацеливающий пептид (MTP), предпочтительно, в котором MTP представляет собой N-концевую область ND.
В другом варианте осуществления, ND способен расщепляться внутри MTP или сразу после MTP с образованием процессированного полипептида NifD (CND) в ходе экспрессии экзогенного полинуклеотида в растительной клетке, при этом CND содержит аминокислотную последовательность в своей N-концевой области (последовательность scar) из C-концевой аминокислоты MTP или не содержит последовательности scar.
В предпочтительном варианте осуществления, MTP расщепляется в растительной клетке с эффективностью, по меньшей мере, 50%, и/или когда CND присутствует в растительной клетке на более высоком уровне, чем ND, предпочтительно в соотношении, составляющем более чем 2:1, более предпочтительно, составляющем более чем 3:1 или 4:1.
В предпочтительном варианте осуществления, CND имеет функциональность NifD.
В дополнительном или другом варианте осуществления вышеуказанных аспектов, экзогенный полинуклеотид кодирует ND, который представляет собой слитой полипептид (слитой полипептид NifD-линкер-NifK), содержащий, в определенном порядке: аминокислотную последовательность NifD, аминокислотную последовательность линкера (линкер) и аминокислотную последовательность полипептида NifK (NK), где аминокислотная последовательность линкера имеет длину в 8-50 остатков, предпочтительно около 30 остатков, которая трансляционно слита с ND и NK. В предпочтительном варианте осуществления, ND дополнительно содержит митохондриальный нацеливающий пептид (MTP), где MTP трансляционно слит в N-концевой области аминокислотной последовательности NifD. В наиболее предпочтительном варианте осуществления, ND способен расщепляться внутри MTP или сразу после MTP с образованием процессированного полипептида NifD (CND) в ходе экспрессии экзогенного полинуклеотида в растительной клетке, при этом CND содержит в своей N-концевой области последовательность scar или не содержит последовательности scar.
В варианте осуществления вышеуказанных аспектов, ND или CND имеет функциональность NifD, или ND (полипептид NifD-линкер-NifK) имеет функциональность как NifD, так и NifK. В некоторых вариантах осуществления, полипептид NifD представляет собой полипептид AnfD, а полипептид NifK представляет собой полипептид AnfK.
В некоторых вариантах осуществления вышеупомянутых аспектов, MTP содержит любой из MTP, раскрытый в данном документе, например, MTP содержит в длину около 51 аминокислоты из MTP γ-субъединицы F1-ATPase.
В некоторых вариантах осуществления, CND содержит последовательность scar длиной от 1 до 45 аминокислот, предпочтительно от 1 до 20 аминокислот, более предпочтительно от 1 до 10 или 11-20 аминокислот, трансляционно слитую в N-концевой области аминокислотной последовательности NifD.
В дополнительном или другом варианте осуществления, ND или CND, или оба, например полипептид NifD-линкер-NifK, находятся в митохондриях растительной клетки, предпочтительно в митохондриальном матриксе (MM) растительной клетки.
В другом варианте осуществления ND или CND, или оба, например полипептид NifD-линкер-NifK, преимущественно растворимы в митохондриях растения. Предпочтительно, чтобы, по меньшей мере, 60% или, по меньшей мере, 75% CND, находящегося в митохондриях растения, было растворимо. Степень растворимости предпочтительно определяют описанным в примерах способом.
В дополнительном или другом варианте осуществления, ND, например полипептид NifD-линкер-NifK, содержит аминокислоту, отличную от тирозина (Y), в позиции, соответствующей аминокислоте 100 из SEQ ID NO:18.
В одном из вариантов осуществления, ND, например полипептид NifD-линкер-NifK, содержит глутамин (Q) или лизин (K) в позиции, соответствующей аминокислоте 100 из SEQ ID NO:18, или лейцин (L), или метионин (M), или фенилаланин (F) в позиции, соответствующей аминокислоте 100 в SEQ ID NO:18.
В другом варианте осуществления, ND содержит Q, K, L, или M в позиции, соответствующей аминокислоте 100 из SEQ ID NO:18.
В другом варианте осуществления, ND содержит L или M в позиции, соответствующей аминокислоте 100 из SEQ ID NO:18.
В другом варианте осуществления, ND содержит Q, K или L в позиции, соответствующей аминокислоте 100 из SEQ ID NO:18.
В другом варианте осуществления, ND содержит Q, K или M в позиции, соответствующей аминокислоте 100 из SEQ ID NO:18.
В другом варианте осуществления, ND содержит Q, K или F в позиции, соответствующей аминокислоте 100 из ID NO:18.
В дополнительном или другом варианте осуществления, ND, например полипептид NifD-линкер-NifK, содержит последовательность RRNX (SEQ ID NO:154) в позиции, соответствующей аминокислотам 97-100 из SEQ ID NO:18, где Х представляет собой любую аминокислоту отличную от Y.
В некоторых вариантах осуществления, X представляет собой Q или K, или L, M или F, или L или M, или Q, K или L, или Q, K или M, или Q, K, или F.
В дополнительном варианте осуществления, растительная клетка содержит один или несколько экзогенных полинуклеотидов, предпочтительно от 2 до 8 экзогенных полинуклеотидов, которые кодируют один или несколько слитых полипептидов Nif (NF), отличных от ND, где каждый NF содержит MTP в N-концевой области NF, и (ii) полипептидную последовательность Nif (NP), где каждый MTP независимо является одинаковым или различным, и каждый NP независимо является одинаковым или различным.
В одном из вариантов осуществления, каждый NF способен расщепляться внутри своего MTP или сразу после MTP с образованием процессированного полипептида Nif (CNF), когда один или несколько экзогенных полинуклеотидов экспрессируются в растительной клетке, при этом каждый CNF либо содержит в своей N-концевой области последовательность scar, либо не содержит последовательность scar.
В одном из вариантов осуществления, по меньшей мере, один из полипептидов NF представляет собой полипептид NifK или полипептид NifH, или оба полипептида NifK и NifH.
В дополнительном или другом варианте осуществления, растительная клетка содержит аминокислотную последовательность NK, где С-конец полипептида представляет собой С-конец NifK дикого типа, т. е. у NK отсутствует какое-либо искусственно добавленное С-концевое продолжение.
В дополнительном или другом варианте осуществления вышеуказанных аспектов, экзогенный полинуклеотид кодирует слитой полипептид NifE-линкер-NifN (NifE-линкер-NifN), содержащий, в определенном порядке, аминокислотную последовательность NifE (NE), аминокислотную последовательность линкера (линкер) и аминокислотную последовательность полипептида NifN (NN), где аминокислотная последовательность линкера имеет длину в 20-70 остатков, предпочтительно около 46 остатков, которая трансляционно слита с NE и NN. В предпочтительном варианте осуществления, полипептид NifE-линкер-NifN содержит митохондриальный нацеливающий пептид (MTP), где MTP трансляционно слит в N-концевой области аминокислотной последовательности NE. В наиболее предпочтительном варианте осуществления, полипептид NifE-линкер-NifN способен расщепляться внутри MTP или сразу после MTP с образованием процессированного полипептида NifD (CNE) в ходе экспрессии экзогенного полинуклеотида в растительной клетке, при этом CNE содержит в своей N-концевой области последовательность scar или не содержит последовательности scar.
В дополнительном или другом варианте осуществления линкер полипептида NifE-линкер-NifN состоит, по меньшей мере, из 30 аминокислот, или, по меньшей мере, около из 40 аминокислот, или от около 20 аминокислот до около 60 аминокислот, или от около 30 аминокислот до около 70 аминокислот, или от около 30 аминокислот до около 60 аминокислот, или от около 30 аминокислот до около 50 аминокислот, или около 25 аминокислот, или около 30 аминокислот, или около 35 аминокислот, или около 40 аминокислот или около 45 аминокислот, или около 46 аминокислот, или около 50 аминокислот, или около 55 аминокислот в длину. Наиболее предпочтительно линкер имеет около 30 аминокислот в длину для слитого полипептида NifD-линкер-NifK и около 46 аминокислот в длину для слитого полипептида NifE-линкер-NifN. В контексте данного документы термин «около 30» означает 27, 28, 29, 30, 31, 32 или 33 аминокислоты, а термин «около 46» означает 41, 42, 43, 44, 45, 46, 47, 48, 49, 50 или 51 аминокислоту.
В дополнительном или другом варианте осуществления, линкер имеет достаточную длину, позволяя ND и NK или NE и NN связываться в функциональной конфигурации в растительной или бактериальной клетке. В одном из вариантов осуществления, линкер имеет в длину от 8 до 50 аминокислот. Предпочтительно, линкер состоит из, по меньшей мере, из около 20 аминокислот, по меньшей мере, из около 25 аминокислот или, по меньшей мере, из около 30 аминокислот в длину. Более предпочтительно, линкер для слитого полипептида NifD-линкер-NifK имеет в длину от 25 до 35 аминокислот.
В дополнительном или другом варианте осуществления слитой полипептид способен расщепляться внутри своего MTP или сразу после MTP с образованием процессированного полипептида (CDK), когда экзогенный полинуклеотид экспрессируется в растительной клетке, при этом CDK содержит, по порядку, необязательно, последовательность scar, аминокислотную последовательность NifD, аминокислотную последовательность линкера и аминокислотную последовательность NK. Если расщепление происходит сразу после MTP, то пептид scar отсутствует.
В некоторых вариантах осуществления, растительная клетка содержит слитой полипептид, CDK или оба.
В дополнительном или другом варианте осуществления, CDK содержит последовательность scar длиной от 1 до 45 аминокислот, предпочтительно от 1 до 20 аминокислот, более предпочтительно от 1 до 10 или 11-20 аминокислот, трансляционно слитую в N-концевой области аминокислотной последовательности NifD.
В дополнительном или другом варианте осуществления, CDK обладает функциональностью и NifD и NifK.
В дополнительном варианте осуществления, растительная клетка содержит один или несколько экзогенных полинуклеотидов которые кодируют один или несколько полипептидов Nif (NF), отличных от ND и NK, где каждый NF содержит (i) MTP в N-концевой области NF, и (ii) полипептидную последовательность Nif (NP), где каждый MTP независимо является одинаковым или различным, и каждый NP независимо является одинаковым или различным.
В дополнительном или другом варианте осуществления, каждый NF способен расщепляться внутри своего MTP или сразу после MTP с образованием процессированного полипептида Nif (CNF), когда один или несколько экзогенных полинуклеотидов экспрессируются в растительной клетке, при этом каждый CNF либо содержит в своей N-концевой области последовательность scar, либо не содержит последовательность scar.
В одном из вариантов осуществления, по меньшей мере, один из полипептидов NF представляет собой полипептид NifH.
В варианте осуществления любого из вышеуказанных аспектов, растительная клетка содержит экзогенные полинуклеотиды, кодирующие полипептиды Nif, содержащие (i) полипептиды NifD, NifH, NifK, NifB, NifE и NifN, предпочтительно в митохондриальном матриксе растительной клетки.
В дополнительном или другом варианте осуществления любого из вышеуказанных аспектов каждый MTP содержит, по меньшей мере, 10 аминокислот, предпочтительно имеет длину, составляющую от 10 до 80 аминокислот.
В дополнительном или другом варианте осуществления любого из вышеуказанных аспектов MTP, или, по меньшей мере, один MTP, или все MTP независимо содержат MTP предшественника митохондриального белка или его варианта, предпочтительно растительного MTP.
В дополнительном или другом варианте осуществления любого из вышеуказанных аспектов, один или несколько или все экзогенные полинуклеотиды интегрируются в ядерный геном клетки, предпочтительно в виде сцепленной последовательности нуклеиновой кислоты и/или экспрессируются в ядре клетки.
В варианте осуществления любого из вышеуказанных аспектов, клетка представляет собой клетку, отличную от протопласта Arabidopsis thaliana, или клетку, отличную от клетки Nicotiana benthamiana.
Авторы настоящего изобретения также продуцировали растительные клетки, которые продуцируют комбинации полипептидов Nif, которые, по меньшей мере, частично растворимы в митохондриях растения.
Таким образом, в одном аспекте настоящее изобретение относится к растительной клетке, содержащей митохондрии и, по меньшей мере, 3, по меньшей мере, 4, по меньшей мере, 5, по меньшей мере, 6, по меньшей мере, 7, по меньшей мере, 8, по меньшей мере, 9, по меньшей мере, 10 или, по меньшей мере, 11 полипептидов Nif, где полипептиды Nif выбираются из группы, состоящей из NifF, NifM, NifN, NifS, NifU, NifW, NifY, NifZ, NifV, NifH и NifD-NifK, и где каждый из, по меньшей мере, 3, по меньшей мере, 4, по меньшей мере, 5, по меньшей мере, 6, по меньшей мере, 7, по меньшей мере, 8, по меньшей мере, 9, по меньшей мере, 10 или, по меньшей мере, 11 полипептидов Nif, по меньшей мере, частично растворим в митохондриях.
В одном из вариантов осуществления, растительная клетка содержит полипептид NifV. Предпочтительно NifV продуцирует гомоцитрат. Более предпочтительно, полипептид NifV, по меньшей мере, частично растворим в митохондриях растительной клетки. В одном из вариантов осуществления, полипептид NifV представляет собой NifV согласно изобретению.
В другом варианте осуществления растительная клетка содержит, по меньшей мере, полипептиды NifS, NifU или оба NifS и NifU и, необязательно, полипептиды NifV.
В другом варианте осуществления, растительная клетка содержит, по меньшей мере, полипептиды NifH, NifM или оба NifH и NifM и, необязательно, один или несколько или все NifV, NifS и NifU.
В другом варианте осуществления, растительная клетка содержит полипептиды NifF, NifH или NifD-NifK, или NifH и NifD-NifK, или NifF, NifH и NifD-NifK, и, необязательно, один или несколько или все из полипептидов NifV, NifS, NifU, NifH и NifM.
В одном из вариантов осуществления, полипептид NifD представляет собой полипептид AnfD, полипептид NifH представляет собой полипептид AnfH, а полипептид NifD-NifK представляет собой полипептид AnfD-AnfK. В предпочтительном варианте осуществления, растительная клетка дополнительно содержит полипептид AnfG, который, по меньшей мере, частично растворим в митохондриях.
В одном из вариантов осуществления, каждый из, по меньшей мере, 3, по меньшей мере, 4, по меньшей мере, 5, по меньшей мере, 6, по меньшей мере, 7, по меньшей мере, 8, по меньшей мере, 9, по меньшей мере, 10 или, по меньшей мере, 11 полипептидов Nif после расщепления MPP, независимо, по меньшей мере, до 10%, по меньшей мере, до 20%, по меньшей мере, до 30%, по меньшей мере, до 40% или, по меньшей мере, до 50% растворимы в митохондриях. Полипептиды Nif до 80% или до 90%, или даже полностью растворимы в митохондриях растительной клетки.
В одном из вариантов осуществления, по меньшей мере, 3, по меньшей мере, 4, по меньшей мере, 5, по меньшей мере, 6, по меньшей мере, 7, по меньшей мере, 8, по меньшей мере, 9, по меньшей мере, 10 или, по меньшей мере, 11 полипептидов Nif, каждый независимо, содержит митохондриальный нацеливающий пептид (MTP), или C-концевой пептид, полученный в результате расщепления MTP, или комбинацию как MPP-процессированных, так и непроцессированных форм, предпочтительно, где MTP находится на N-конце каждой из, по меньшей мере, 3, по меньшей мере, 4, по меньшей мере, 5, по меньшей мере, 6, по меньшей мере, 7, по меньшей мере, 8, по меньшей мере, 9, по меньшей мере, 10 или по меньшей мере, 11 полипептидов Nif, или MPP-процессированная форма не имеет C-концевого пептида на N-конце полипептида Nif.
В одном из вариантов осуществления, каждый MTP, независимо, расщепляется в растительной клетке с эффективностью, по меньшей мере, 50%, и/или где каждый из, по меньшей мере, 3, по меньшей мере, 4, по меньшей мере, 5, по меньшей мере, 6, по меньшей мере, 7, по меньшей мере, 8, по меньшей мере, 9, по меньшей мере, 10 или, по меньшей мере, 11 процессированных полипептидов Nif, независимо, присутствуют в растительной клетке на более высоком уровне, чем соответствующий полипептид Nif, предпочтительно, в соотношении более чем 1:1, более чем 2:1, более чем 3:1 или более чем 4: 1.
В одном из вариантов осуществления, растительная клетка содержит слитой полипептид NifD-линкер-NifK, содержащий, в определенном порядке, аминокислотную последовательность NifD (ND), аминокислотную последовательность линкера и аминокислотную последовательность полипептида NifK (NK), где линкерная аминокислотная последовательность имеет длину в 8-50 остатков, предпочтительно в 16-50 остатков, более предпочтительно около 26 или около 30 остатков или наиболее предпочтительно 26 или 30 остатков, которая трансляционно слита с ND и NK.
В дополнительном варианте осуществления, слитой полипептид NifD-линкер-NifK содержит митохондриальный нацеливающий пептид (MTP) или C-концевой пептид, полученный в результате расщепления MTP, или присутствует комбинация как MPP-процессированных, так и непроцессированных форм, где MTP трансляционно слит в N-концевой области слитого полипептида NifD-NifK.
В одном из вариантов осуществления, по меньшей мере, 3, по меньшей мере, 4, по меньшей мере, 5, по меньшей мере, 6, по меньшей мере, 7, по меньшей мере, 8, по меньшей мере, 9, по меньшей мере, 10 или, по меньшей мере, 11 процессированных полипептидов Nif, каждый независимо содержит С-концевой пептид, полученный в результате расщепления MTP длиной от 1 до 45 аминокислот, предпочтительно от 1 до 20 аминокислот, более предпочтительно, от 1 до 10 или 11-20 аминокислот, трансляционно слитых в N-концевой области полипептида Nif.
В одном из вариантов осуществления, по меньшей мере, 3, по меньшей мере, 4, по меньшей мере, 5, по меньшей мере, 6, по меньшей мере, 7, по меньшей мере, 8, по меньшей мере, 9, по меньшей мере, 10 или, по меньшей мере, 11 полипептидов Nif, или, по меньшей мере, 3, по меньшей мере, 4, по меньшей мере, 5, по меньшей мере, 6, по меньшей мере, 7, по меньшей мере, 8, по меньшей мере, 9, по меньшей мере, 10 или, по меньшей мере, 11 процессированных полипептидов Nif являются функциональными полипептидами Nif.
В одном из вариантов осуществления, по меньшей мере 3, по меньшей мере, 4, по меньшей мере, 5, по меньшей мере, 6, по меньшей мере, 7, по меньшей мере, 8, по меньшей мере, 9, по меньшей мере, 10 или, по меньшей мере, 11 полипептидов Nif или, предпочтительно, по меньшей мере, 3, по меньшей мере, 4, по меньшей мере, 5, по меньшей мере, 6, по меньшей мере, 7, по меньшей мере, 8, по меньшей мере, 9, по меньшей мере, 10 или, по меньшей мере, 11 процессированных полипептидов Nif, находятся в митохондриях растительной клетки, предпочтительно в митохондриальном матриксе (ММ) растительной клетки.
В одном из вариантов осуществления, по меньшей мере, 3, по меньшей мере, 4, по меньшей мере, 5, по меньшей мере, 6, по меньшей мере, 7, по меньшей мере, 8, по меньшей мере, 9, по меньшей мере, 10 или, по меньшей мере, 11 полипептидов Nif или, предпочтительно, по меньшей мере, 3, по меньшей мере, 4, по меньшей мере, 5, по меньшей мере, 6, по меньшей мере, 7, по меньшей мере, 8, по меньшей мере, 9, по меньшей мере, 10 или, по меньшей мере, 11 процессированных полипептидов Nif, или оба, независимо друг от друга, преимущественно растворимы в митохондриях растения (т. е. растворимы в митохондриях более чем на 50%). Процессированные полипептиды Nif растворимы, предпочтительно, до 80% или до 90% или даже полностью в митохондриях растительной клетки. Растворимость полипептида можно определить, как описано в данном документе.
В одном из вариантов осуществления, слитой полипептид NifD или слитой полипептид NifD-линкер-NifK, или их MPP-расщепленные продукты присутствуют в растительной клетке и (а) устойчивы к расщеплению протеазой на сайте в пределах аминокислотной последовательности, соответствующих аминокислот 97-100 из SEQ ID NO:18 и/или (b) содержат аминокислотную последовательность, отличную от RRNY (SEQ ID NO:101), в позициях, соответствующих аминокислотам 97-100 из SEQ ID NO:18. В одном из вариантов осуществления, ND содержит аминокислоту, отличную от тирозина (Y), в позиции, соответствующей аминокислоте 100 из SEQ ID NO:18. В одном из вариантов осуществления, ND содержит глутамин (Q) или лизин (K) в позиции, соответствующей аминокислоте 100 из SEQ ID NO:18, или лейцин (L), или метионин (M), или фенилаланин (F) в позиции, соответствующей аминокислоте 100 из SEQ ID NO:18.
В одном из вариантов осуществления, MTP имеет в длину около 51 аминокислоты из MTP γ-субъединицы F1-ATPase.
В одном из вариантов осуществления, растительная клетка содержит аминокислотную последовательность NK, где С-конец полипептида представляет собой С-конец NifK дикого типа.
В одном из вариантов осуществления, линкер представляет собой, по меньшей мере, около 20 аминокислот, или, по меньшей мере, около 30 аминокислот, или по меньшей мере, около 40 аминокислот, или от около 20 аминокислот до около 70 аминокислот, или от около 30 аминокислот до около 70 аминокислот, или от около 30 аминокислот до около 60 аминокислот, или от около 30 аминокислот до около 50 аминокислот, или около 25 аминокислот, или около 30 аминокислот, или около 35 аминокислот, или около 40 аминокислот, или около 45 аминокислот, или около 46 аминокислот, или около 50 аминокислот, или около 55 аминокислот в длину.
В одном из вариантов осуществления, слитой полипептид NifD-линкер-NifK способен расщепляться внутри своего MTP или сразу после MTP с образованием процессированного полипептида (CDK), при этом CDK содержит, по порядку, необязательно, C-концевой пептид, полученный в результате расщепления MTP, аминокислотную последовательность NifD (ND), аминокислотную последовательность линкера и аминокислотную последовательность NK.
В одном из вариантов осуществления, растительная клетка дополнительно содержит слитой полипептид или CDK, или оба.
В одном из вариантов осуществления, CDK содержит последовательность scar длиной от 1 до 45 аминокислот, предпочтительно от 1 до 20 аминокислот, более предпочтительно от 1 до 10 или 11-20 аминокислот, трансляционно слитую в N-концевой области аминокислотной последовательности NifD.
В одном из вариантов осуществления, CDK обладает функциональностью и NifD, и NifK.
В одном из вариантов осуществления, ND представляет собой AnfD, а NK представляет собой AnfK.
В одном из вариантов осуществления, MTP имеет в длину около 51 аминокислоты из MTP γ-субъединицы F1-ATPase.
В одном из вариантов осуществления, каждый MTP содержит, по меньшей мере, 10 аминокислот, предпочтительно имеет длину, составляющую от 10 до 80 аминокислот.
В одном из вариантов осуществления, указанный MTP, или, по меньшей мере, один MTP, или все MTP независимо содержат MTP предшественника митохондриального белка или его варианта, предпочтительно растительного MTP.
В одном из вариантов осуществления, по меньшей мере 3, по меньшей мере, 4, по меньшей мере, 5, по меньшей мере, 6, по меньшей мере, 7, по меньшей мере, 8, по меньшей мере, 9, по меньшей мере, 10 или, по меньшей мере, 11 полипептидов Nif кодируются, по меньшей мере, 3, по меньшей мере, 4, по меньшей мере, 5, по меньшей мере, 6, по меньшей мере, 7, по меньшей мере, 8, по меньшей мере, 9, по меньшей мере, 10 или, по меньшей мере, 11 экзогенными полинуклеотидами, из которых, по меньшей мере, 3, по меньшей мере, 4, по меньшей мере, 5, по меньшей мере, 6, по меньшей мере, 7, по меньшей мере, 8, по меньшей мере, 9, по меньшей мере, 10 или, по меньшей мере, 11 интегрируются в ядерный геном клетки, предпочтительно в виде сцепленной последовательности нуклеиновой кислоты и/или экспрессируются в ядре растительной клетки.
В варианте осуществления любого из вышеуказанных аспектов, клетка представляет собой клетку, отличную от протопласта Arabidopsis thaliana, или клетку, отличную от клетки Nicotiana benthamiana.
Авторы настоящего изобретения также успешно экспрессировали в митохондриях растения комбинацию полипептидов Nif, необходимую для минимального комплекса нитрогеназы.
Таким образом, в другом аспекте настоящее изобретение относится к растительной клетке, содержащей митохондрии и экзогенные полинуклеотиды, которые кодируют, по меньшей мере, 8 или, по меньшей мере, 9 слитых полипептидов Nif, причем каждый экзогенный полинуклеотид содержит промотор, который функционально связан с нуклеотидной последовательностью, кодирующей один из слитых полипептидов Nif, и который экспрессирует нуклеотидную последовательность в растительной клетке, причем каждый слитой полипептид Nif, независимо, содержит митохондриально-нацеливающий пептид (MTP), где слитые полипептиды Nif содержат: (i) слитые полипептиды NifH, NifB, NifF, NifJ, NifS, NifU и NifV; и либо (ii) слитой полипептид NifD и слитой полипептид NifK; либо (iii) слитой полипептид NifD-линкер-NifK, который содержит: последовательность NifD, имеющую С-конец; олигопептидный линкер; и последовательность NifK, имеющую N-конец, где олигопептидный линкер трансляционно слит с C-концом последовательности NifD и N-концом последовательности NifK, где продукты, расщепленные митохондриальной процессинговой протеазой (MPP), по меньшей мере, слитых полипептидов NifH, NifF, NifS и NifU, по меньшей мере, каждый частично растворим в митохондриях растительной клетки, или MPP- расщепленный продукт слитых полипептидов NifD и NifK из (ii), если они присутствуют в растительной клетке, по меньшей мере, частично растворимы в митохондриях растительной клетки, или MПП-расщепленный продукт слитого полипептида NifD-линкер-NifK из (iii), если он присутствует в растительной клетке, по меньшей мере, частично растворим в митохондриях растительной клетки, и где слитой полипептид NifV и/или его МРР-расщепленный продукт, продуцирует гомоцитрат в растительной клетке и, по меньшей мере, частично растворим в митохондриях растительной клетки.
В другом аспекте настоящее изобретение относится к растительной клетке, содержащей митохондрии и экзогенные полинуклеотиды, которые кодируют, по меньшей мере, 2, по меньшей мере, 3, по меньшей мере, 4, по меньшей мере, 5 или, по меньшей мере, 6 слитых полипептидов Nif, причем каждый экзогенный полинуклеотид содержит промотор, который функционально связан с нуклеотидной последовательностью, которая кодирует один из слитых полипептидов Nif и которая экспрессирует нуклеотидную последовательность в растительной клетке, причем каждый слитой полипептид Nif независимо содержит митохондриальный нацеливающий пептид (MTP), где слитые полипептиды Nif содержат: (i) один или более чем один или все слитые полипептиды NifW, NifX, NifY и NifZ; и либо (ii) слитой полипептид NifD и слитой полипептид NifK; либо (iii) слитой полипептид NifD-линкер-NifK, который содержит: последовательность NifD, имеющую С-конец; олигопептидный линкер; и последовательность NifK, имеющую N-конец; где олигопептидный линкер трансляционно слит с С-концом последовательности NifD и N-концом последовательности NifK, где продукты, расщепленные митохондриальной процессинговой протеазой (MPP), по меньшей мере, слитых полипептидов NifW, NifX, NifY и NifZ, если они присутствуют в растительной клетке, каждый, по меньшей мере, частично растворим в митохондриях растительной клетки, где либо MPP-расщепленные продукты слитых полипептидов NifD и NifK из (ii), если они присутствуют в растительной клетке, по меньшей мере, частично растворимы в митохондриях растительной клетки, либо MPP-расщепленный продукт слитого полипептида NifD-линкер-NifK из (iii), присутствует в растительной клетке в большем количестве, чем количество MPP-расщепленных продуктов слитого полипептида NifD и слитого полипептида NifK или MPP-расщепленного продукта слитого полипептида NifD-линкер-NifK, если он присутствует в соответствующей растительной клетке, лишенной экзогенных полинуклеотидов, кодирующих один или несколько или все слитые полипептиды NifW, NifX, NifY и NifZ из (i).
В другом аспекте настоящее изобретение относится к растительной клетке, содержащей митохондрии и экзогенные полинуклеотиды, которые кодируют, по меньшей мере, 5, по меньшей мере, 6, по меньшей мере, 7, по меньшей мере, 8 или, по меньшей мере, 9 слитых полипептидов Nif, причем каждый экзогенный полинуклеотид содержит промотор, который функционально связан с нуклеотидной последовательностью, которая кодирует один из слитых полипептидов Nif, и которая экспрессирует нуклеотидную последовательность в растительной клетке, где каждый слитой полипептид Nif независимо содержит митохондриальный нацеливающий пептид (MTP), где слитые полипептиды Nif содержат (i) слитые полипептиды NifH, NifS и NifU и, необязательно, полипептид NifM, (ii) один или несколько или все слитые полипептиды NifW, NifX, NifY и NifZ и либо (iii) слитой полипептид NifD и слитой полипептид NifK, или (iv) слитой полипептид NifD-линкер-NifK, который содержит последовательность NifD, имеющую С-конец, олигопептидный линкер и последовательность NifK, имеющую N-конец, где олигопептидный линкер трансляционно слит с С-концом последовательности NifD и N-концом последовательности NifK, где продукты, расщепленные митохондриальной процессинговой протеазой (MPP) слитых полипептидов NifS и NifU, по меньшей мере, частично растворимы в митохондриях растительной клетки, где MPP-расщепленные продукты слитых полипептидов NifW, NifX, NifY и NifZ, если они присутствуют в растительной клетке, по меньшей мере, частично растворимы в митохондриях растительной клетки, где MPP-расщепленные продукты слитых полипептидов NifD и NifK из (iii), если они присутствуют в растительной клетке, по меньшей мере, частично растворимы в митохондриях растительной клетки, где MPP-расщепленный продукт слитого полипептида NifD-линкер-NifK из (iv), если он присутствует в растительной клетке, по меньшей мере, частично растворим в митохондриях растительной клетки, и где либо МРР-расщепленные продукты слитого полипептида NifD и слитого полипептида NifK из iii), либо МРР-расщепленный продукт слитого полипептида NifD-линкер-NifK из iv) присутствуют в растительной клетке в виде комплекса с Р-кластером.
В одном из вариантов осуществления, растительная клетка содержит слитой полипептид NifH, который представляет собой слитой полипептид AnfH, где слитой полипептид NifD, если он присутствует, является слитым полипептидом AnfD, слитой полипептид NifK, если он присутствует, представляет собой слитой полипептид AnfK, слитой полипептид NifD-линкер-NifK, если присутствует, представляет собой слитой полипептид AnfD-линкер-AnfK, а растительная клетка дополнительно содержит экзогенный полинуклеотид, который кодирует слитой полипептид AnfG, который содержит MTP, где экзогенный полинуклеотид, который кодирует слитой полипептид AnfG, содержит промотор, который функционально связан с нуклеотидной последовательностью, которая кодирует слитой полипептид AnfG и который экспрессирует указанную нуклеотидную последовательность в растительной клетке, и где MPP-расщепленный продукт слитого полипептида AnfG, по меньшей мере, частично растворим в митохондриях растительной клетки.
В варианте осуществления трех вышеупомянутых аспектов, слитой полипептид NifD или слитой полипептид NifD-линкер-NifK присутствует в растительной клетке и (а) устойчив к расщеплению протеазой на сайте в аминокислотной последовательности, соответствующей аминокислотам 97 -100 из SEQ ID NO:18 и/или (b) содержит аминокислотную последовательность, отличную от RRNY (SEQ ID NO:101) в позициях, соответствующих аминокислотам 97-100 из SEQ ID NO:18.
Насколько им известно, авторы настоящего изобретения первыми создали растительную клетку, содержащую полипептид NifV, который, по меньшей мере, частично растворим в митохондриях. Таким образом, в другом аспекте настоящее изобретение относится к растительной клетке, содержащей полипептид NifV (NV), где NV, по меньшей мере, частично растворим в митохондриях растительной клетки, предпочтительно в ММ растительной клетки.
В одном из вариантов осуществления, NV способен продуцировать или продуцирует гомоцитрат в клетке.
В одном из вариантов осуществления, полипептид NV включает аминокислоты, имеющие последовательность, представленную как любую из SEQ ID NO: 163, 206-209, 211 или 212, как его биологически активный фрагмент или имеет аминокислотную последовательность, которая идентична, по меньшей мере, на 30%, идентична, по меньшей мере, на 40% идентична, по меньшей мере, на 50% идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80% идентична, по меньшей мере, на 90% идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99% тому, что представлено в любой одной или нескольких SEQ ID NO:163, 206-209, 211 или 212, и способно продуцировать гомоцитрат в клетке.
В одном из вариантов осуществления этого аспекта, настоящее изобретение относится к растительной клетке, содержащей митохондрии и экзогенный полинуклеотид, который кодирует полипептид NifV (NV), причем экзогенный полинуклеотид содержит промотор, который функционально связан с нуклеотидной последовательностью, кодирующей NV, и которая экспрессирует указанную нуклеотидную последовательность в растительной клетке, где NV продуцирует гомоцитрат в растительной клетке и, по меньшей мере, частично растворим в митохондриях растительной клетки, причем экзогенный полинуклеотид, предпочтительно, интегрирован в ядерный геном растительной клетки и/или экспрессируется в ядре растительной клетки и, необязательно, где NV содержит митохондриальный нацеливающий пептид (MTP).
В другом аспекте, настоящее изобретение относится к растительной клетке, содержащей экзогенный полинуклеотид, который кодирует полипептид NifD (ND), который: (а) устойчив к расщеплению протеазой на сайте в аминокислотной последовательности, соответствующей аминокислотам 97-100 из SEQ ID NO:18; и/или (b) содержит аминокислотную последовательность, отличающуюся от RRNY (SEQ ID NO:101) в позициях, соответствующих аминокислотам 97-100 из SEQ ID NO:18, где экзогенный полинуклеотид содержит промотор, который функционально связан с нуклеотидной последовательностью, которая кодирует ND и которая экспрессирует указанную нуклеотидную последовательность в растительной клетке, и где полипептид NifD предпочтительно содержит MTP.
В одном из вариантов осуществления, растительная клетка содержит экзогенный полинуклеотид, кодирующий полипептид NifK (NK), где экзогенный полинуклеотид, кодирующий NK, содержит промотор, который функционально связан с нуклеотидной последовательностью, кодирующей NK и который экспрессирует указанную нуклеотидную последовательность в растительной клетке, где ND имеет С-конец, а NK имеет N-конец, и где либо (i) NK содержит митохондриальный нацеливающий пептид (MTP), либо (ii) ND и NK трансляционно слиты в виде слитого полипептида NifD-линкер-NifK, который содержит олигопептидный линкер, где олигопептидный линкер трансляционно слит с C-концом ND и N-концом NK.
В одном из вариантов осуществления, растительная клетка содержит экзогенный полинуклеотид, который кодирует слитой полипептид NifH (NH), где экзогенный полинуклеотид, кодирующий NH, содержит промотор, который функционально связан с нуклеотидной последовательностью, кодирующей NH и который экспрессирует указанную нуклеотидную последовательность в растительной клетке, где NH включает митохондриальный нацеливающий пептид (MTP), и, предпочтительно, где NH и/или его MPP-расщепленный продукт, по меньшей мере, частично растворим в митохондриях растительной клетки.
В одном из вариантов осуществления, MPP-расщепленный продукт, по меньшей мере, одного или нескольких, или, предпочтительно, всех слитых полипептидов Nif, по меньшей мере, частично растворим в митохондриях растительной клетки, предпочтительно, где MPP-расщепленный продукт каждого из слитых полипептидов NifD, NifK и NifD-линкер-NifK, если они присутствуют в растительной клетке, и полипептида NifH, по меньшей мере, частично растворим в митохондриях растительной клетки.
Насколько им известно, авторы настоящего изобретения первыми создали растительную клетку, содержащую полипептид NifH, который, по меньшей мере, частично растворим в митохондриях. Таким образом, в другом аспекте настоящее изобретение относится к растительной клетке, содержащей полипептид NifH (NH), где NH, по меньшей мере, частично растворим в митохондриях.
В одном из вариантов осуществления, NH кодируется экзогенным полинуклеотидом, который интегрирован в ядерный геном клетки, предпочтительно, в виде сцепленной последовательности нуклеиновой кислоты с экзогенными полинуклеотидами, кодирующими слитые полипептиды NifD, NifK и NifD-линкер-NifK, при наличии в растительной клетке.
В другом аспекте настоящее изобретение относится к растительной клетке, содержащей экзогенный полинуклеотид, который кодирует слитой полипептид NifH (NH), где экзогенный полинуклеотид содержит промотор, который функционально связан с нуклеотидной последовательностью, кодирующей NH и экспрессирующей указанную нуклеотидную последовательность в растительной клетке, где NH включает митохондриальный нацеливающий пептид (MTP), где MPP-расщепленный продукт NH, по меньшей мере, частично растворим в митохондриях растительной клетки, и, необязательно, где экзогенный полинуклеотид интегрирован в ядерный геном растительной клетки и/или экспрессируется в ядре растительной клетки.
В некоторых вариантах осуществления каждого из вышеуказанных аспектов, растительная клетка дополнительно содержит экзогенный полинуклеотид, кодирующий полипептид NifM (NM), где экзогенный полинуклеотид, кодирующий NM, содержит промотор, который функционально связан с нуклеотидной последовательностью, кодирующей NM, и который экспрессирует указанную нуклеотидную последовательность в растительной клетке, и где NM необязательно включает митохондриальный нацеливающий пептид (MTP).
В некоторых вариантах осуществления каждого из вышеуказанных аспектов, растительная клетка содержит экзогенные полинуклеотиды, которые кодируют слитые полипептиды NifS и NifU, причем каждый экзогенный полинуклеотид содержит промотор, который функционально связан с нуклеотидной последовательностью, которая кодирует один из слитых полипептидов Nif, и которая экспрессирует нуклеотидную последовательность в растительной клетке, и где каждый слитой полипептид NifS и NifU содержит митохондриальный нацеливающий пептид (MTP).
В некоторых вариантах осуществления каждого из вышеуказанных аспектов, каждый полипептид Nif продуцируется в растительной клетке в виде слитого полипептида Nif, содержащего митохондриальный нацеливающий пептид (MTP), где каждый MTP является, независимо, одинаковым или отличным, предпочтительно, когда MTP находится на N -конце, по меньшей мере, одного или более чем одного или всех слитых полипептидов Nif.
В некоторых вариантах осуществления каждого из вышеуказанных аспектов, каждый слитой полипептид Nif, продуцированный в растительной клетке, независимо расщепляется с помощью MPP либо (i) в последовательности MTP с получением MPP-расщепленного полипептида Nif, при этом MPP-расщепленный полипептид Nif содержит в своей N-концевой области, C-концевой пептид из MTP (пептид scar), либо (ii) сразу после MTP, причем MPP-расщепленный полипептид Nif не содержит C-концевой пептид из MTP.
В некоторых вариантах осуществления каждого из вышеуказанных аспектов, каждый MTP независимо расщепляется в растительной клетке с эффективностью, составляющей, по меньшей мере, 50%, и/или каждый расщепленный полипептид Nif независимо присутствует в растительной клетке на более высоком уровне, чем соответствующий не расщепленный слитой полипептид Nif, предпочтительно, в соотношении более чем 1:1, 2:1 или 3:1.
В некоторых вариантах осуществления каждого из вышеуказанных аспектов, каждый слитой полипептид Nif, по меньшей мере, частично расщепляется в своей последовательности MTP в растительной клетке с продуцированием MPP-расщепленного полипептида Nif, где каждый MPP-расщепленный полипептид Nif независимо содержит пептид (пептид scar) длиной от 1 до 45 аминокислот, предпочтительно длиной от 1 до 20 аминокислот, более предпочтительно диной от 1 до 11 аминокислот или от 11 до 20 аминокислот, полученных из последовательности MTP, трансляционно слитых в N-концевой области MPP-расщепленного полипептида Nif. В некоторых вариантах осуществления, один или несколько пептидов scar, независимо, имеют в длину 2, 3, 4, 5, 6, 7, 8, 9 или 10 аминокислот. В некоторых вариантах осуществления, один или несколько пептидов scar, независимо, имеют в длину 11, 12, 13, 14, 15, 16, 17, 18, 19 или 20 аминокислот или 20-30, 20-40 или 20-50 аминокислот, хотя предпочтительны более короткие последовательности scar. В этих вариантах осуществления, в контексте данного документа, пептид scar содержит любую линкерную последовательность, например, линкер Gly-Gly, используемый в примерах в данном документе, слитой с N-концом последовательности Nif. В одном из вариантов осуществления, последовательность Nif сохраняет Met (начало трансляции Met) из своей последовательности дикого типа на своем N-конце, причем Met не включается в последовательность scar. Кроме того, начало трансляции Met исключается из последовательности Nif. В некоторых вариантах осуществления, дополнительные аминокислоты могут обрезаться с N-конца последовательности Nif, по сравнению с соответствующей последовательностью Nif дикого типа, при условии, что последовательность Nif после обрезки сохраняет функциональность Nif.
В некоторых вариантах осуществления каждого из вышеуказанных аспектов, растительная клетка дополнительно содержит экзогенный полинуклеотид, который кодирует слитой полипептид ферредоксин, предпочтительно слитой полипептид FdxN, где экзогенный полинуклеотид, который кодирует слитой полипептид ферредоксин, содержит промотор, который функционально связан с нуклеотидной последовательностью, которая кодирует слитой полипептид ферредоксин и которая экспрессирует указанную нуклеотидную последовательность в растительной клетке, и где слитой полипептид ферредоксин содержит митохондриальный нацеливающий пептид (MTP).
В одном из вариантов осуществления, MPP-расщепленный продукт слитого полипептида ферредоксина, по меньшей мере, частично растворим в митохондриях растительной клетки, предпочтительно, где экзогенный полинуклеотид интегрирован в ядерный геном растительной клетки и/или экспрессируется в ядре растительной клетки.
В одном из вариантов осуществления, растительная клетка содержит слитой полипептид NifD-линкер-NifK, содержащий, в определенном порядке, аминокислотную последовательность (ND) NifD, олигопептидный линкер и аминокислотную последовательность полипептида NifK (NK), где олигопептидный линкер имеет в длину 8-50 остатков, предпочтительно 16-50 остатков в длину, более предпочтительно около 26 или около 30 остатков в длину, или наиболее предпочтительно 30 остатков в длину, которая трансляционно слита с ND и NK.
В одном из вариантов осуществления, каждый слитой полипептид Nif расщепляется в растительной клетке с образованием полипептида Nif, который представляет собой функциональный полипептид Nif.
В одном из вариантов осуществления, растительная клетка содержит экзогенный полинуклеотид, который кодирует слитой полипептид NifD (ND) или слитой полипептид NifD-линкер-NifK, где ND или слитой полипептид NifD-линкер-NifK содержит аминокислотную последовательность, отличную от RRNY (SEQ ID NO:101) в позициях, соответствующих аминокислотам 97-100 из SEQ ID NO:18, и где ND или слитой полипептид NifD-линкер-NifK, предпочтительно, содержит аминокислоту, отличную от тирозина (Y) в позиции, соответствующей аминокислоте 100 из SEQ ID NO:18.
В одном из вариантов осуществления, ND или слитой полипептид NifD-линкер-NifK, содержит глутамин (Q) или лизин (K) в позиции, соответствующей аминокислоте 100 из SEQ ID NO:18, или лейцин (L), или метионин (M), или фенилаланин (F) в позиции, соответствующей аминокислоте 100 в SEQ ID NO:18.
В одном из вариантов осуществления, растительная клетка содержит экзогенный полинуклеотид, который кодирует слитой полипептид NifK или слитой полипептид NifD-линкер-NifK, где слитой полипептид NifK или слитой полипептид NifD-линкер-NifK имеет С-концевую аминокислотную последовательность, которая аналогична С-концевой аминокислотной последовательности полипептида NifK дикого типа. В некоторых вариантах осуществления, по меньшей мере, две последние, по меньшей мере, три последние, по меньшей мере, четыре последние аминокислоты последовательности являются такими же, как у полипептида NifK дикого типа. Подходящие полипептидные последовательности NifK дикого типа включают SEQ ID NO:3, а также последовательности с Учетными №№: WP_049080161.1, WP_044347163.1, SBM87811.1, WP_047370272.1, WP_014333919.1, WP_012728880.1, WP_011912506.1, WP_065303473.1, WP_018989051.1, prf||2106319A, WP_011021239.1 и другими.
В одном из вариантов осуществления, слитой полипептид NifK или слитой полипептид NifD-линкер-NifK и MPP-расщепленный продукт, имеют аминокислотную последовательность, в которой последние четыре аминокислоты последовательности являются такими же, как последние четыре аминокислоты у полипептида NifK дикого типа.
В одном из вариантов осуществления, аминокислотная последовательность полипептида NifK, по настоящему изобретению, имеет на своем С-конце аминокислоты DLVR (SEQ ID NO:58). В другом варианте осуществления, полипептид NifK имеет на своем С-конце аминокислоты DLIR (SEQ ID NO:239), DVVR (SEQ ID NO:240), DIIR (SEQ ID NO:241), DLTR (SEQ ID NO:242) или INVW (SEQ ID NO:243). В одном из вариантов осуществления, полипептид AnfK имеет на своем С-конце аминокислоты LNVW (SEQ ID NO:244), LNTW (SEQ ID NO:245), LNMW (SEQ ID NO:246), LAMW (SEQ ID NO:247) or LSVW (SEQ ID NO:248).
В некоторых вариантах осуществления, вышеуказанных аспектов, растительная клетка содержит экзогенный полинуклеотид, который кодирует слитой полипептид AnfD-линкер-AnfK, где слитой полипептид AnfD-линкер-AnfK содержит последовательность AnfD, которая имеет С-конец, олигопептидный линкер и последовательность AnfK, которая содержит N-конец, где олигопептидный линкер трансляционно слит с С-концом последовательности AnfD и N-концом последовательности AnfK, при этом олигопептидный линкер имеет в длину, по меньшей мере, около 20 аминокислот, по меньшей мере, около 30 аминокислот, по меньшей мере, около 40 аминокислот, от около 20 аминокислот до около 70 аминокислот, от около 30 аминокислот до около 70 аминокислот, от около 30 аминокислот до около 60 аминокислот, от около 30 аминокислот до около 50 аминокислот, около 25 аминокислот, около 30 аминокислот, около 35 аминокислот, около 40 аминокислот, около 45 аминокислот, около 46 аминокислот, около 50 аминокислот или около 55 аминокислот. Таким образом, в этих вариантах осуществления, последовательность NifD, в вышеупомянутых вариантах осуществления, представляет собой последовательность AnfD, а последовательность NifK представляет собой последовательность AnfK.
В одном из вариантов осуществления, по меньшей мере, один или более чем один, или, предпочтительно, все экзогенные полинуклеотиды интегрируются в ядерный геном растительной клетки и/или экспрессируются в ядре растительной клетки.
В одном из вариантов осуществления, каждый MTP содержит, по меньшей мере, 10 аминокислот, предпочтительно имеет длину, составляющую от 10 до 80 аминокислот.
В одном из вариантов осуществления, по меньшей мере, один из слитых полипептидов Nif содержит MTP, который имеет в длину около 51 аминокислоты из полипептида γ-субъединицы F1-ATPase.
В одном из вариантов осуществления, указанный MTP, или, по меньшей мере, один MTP, или все MTP независимо содержат MTP предшественника митохондриального белка или его варианта, предпочтительно растительного MTP.
В одном из вариантов осуществления, по меньшей мере 3, по меньшей мере, 4, по меньшей мере, 5, по меньшей мере, 6, по меньшей мере, 7, по меньшей мере, 8, по меньшей мере, 9, по меньшей мере, 10 или, по меньшей мере, 11 полипептидов Nif кодируются, по меньшей мере, 3, по меньшей мере, 4, по меньшей мере, 5, по меньшей мере, 6, по меньшей мере, 7, по меньшей мере, 8, по меньшей мере, 9, по меньшей мере, 10 или, по меньшей мере, 11 экзогенными полинуклеотидами, из которых, по меньшей мере, 3, по меньшей мере, 4, по меньшей мере, 5, по меньшей мере, 6, по меньшей мере, 7, по меньшей мере, 8, по меньшей мере, 9, по меньшей мере, 10 или, по меньшей мере, 11 интегрируются в ядерный геном клетки, предпочтительно в виде сцепленной последовательности нуклеиновой кислоты.
В одном из вариантов осуществления вышеуказанных аспектов, клетка не способна давать начало клеткам-потомкам, например, не способна регенерировать культуру клеток или живое растение.
В одном варианте осуществления, растительная клетка, по изобретению, дополнительно определяется одним или несколькими признаками, упомянутыми в данном документе. Предметом рассмотрения является каждая возможная комбинация признаков.
В дополнительном аспекте, настоящее изобретение относится к растению или части растения, органу или ткани, содержащих растительную клетку по изобретению, предпочтительно трансгенному растению или его части, где трансгенное растение или его часть является трансгенным, по меньшей мере, для одного или нескольких экзогенных полинуклеотидов, кодирующих полипептид (ы) Nif.
В одном из вариантов осуществления, часть растения представляет собой семя. В одном варианте осуществления, семя способно прорастать или, в соответствии с другим вариантом, подвергалось процессированию или обработке таким образом, чтобы оно теряло способность прорастать. У клеток семян должна отсутствовать способность к регенерации в культуру клеток или живое растение.
В вариантах осуществления вышеупомянутых аспектов, один или несколько из одного или нескольких экзогенных полинуклеотидов экспрессируются в корнях растения, предпочтительно экспрессируются в верхних частях корней растения, а не в листьях растения. В таких случаях используется промоторная последовательность, которая обеспечивает требуемую тканеспецифическую экспрессию.
В одном из вариантов осуществления, трансгенное растение имеет измененный фенотип по сравнению с соответствующим растением дикого типа, который дает: повышенную урожайность, биомассу, скорость роста, жизненность, прирост азота, обусловленный биологической фиксацией азота, эффективность использования азота, толерантность к абиотическому стрессу и/или толерантность к недостаточности питательных веществ по сравнению с соответствующим растением дикого типа.
В альтернативном варианте осуществления, трансгенное растение имеет такую же скорость роста и/или фенотип, что и соответствующее растение дикого типа.
В вариантах осуществления вышеупомянутых аспектов, растительная клетка, растение или их часть представляет собой клетку злакового растения, растение или его часть, например, пшеницу, рис, кукурузу, тритикале, овес или ячмень, предпочтительно, пшеницу.
В некоторых вариантах осуществления вышеуказанных аспектов, растительная клетка, растение или их часть является гомозиготной или гетерозиготной в одном или нескольких экзогенных полинуклеотидах, предпочтительно гомозиготной во всех экзогенных полинуклеотидах.
В некоторых вариантах осуществления вышеуказанных аспектов, растительная клетка, растение или их часть представляет собой односеменодольную растительную клетку, растение или их часть, например, клетку злакового растения, растение или его часть, например, пшеницу, рис, кукурузу, тритикале, овес или ячмень, предпочтительно пшеницу, или двусемядольную растительную клетку, растение или их часть.
В дополнительном или другом варианте осуществления трансгенное растение растет в поле или часть растения собирали с растения, выращенного в поле. Кроме того, растение выращивали в теплице.
В дополнительном аспекте настоящее изобретение обеспечивает, согласно изобретению, популяцию, по меньшей мере, 100 растений, растущих в поле или в теплице, или части растения, собранные с них.
В другом аспекте настоящее изобретение относится к выделенному или рекомбинантному полипептиду NifD (ND), который устойчив к расщеплению протеазой на сайте в аминокислотной последовательности, соответствующей аминокислотам 97-100 из SEQ ID NO:18.
В дополнительном аспекте настоящее изобретение относится к выделенному или рекомбинантному полипептиду NifD (ND), который содержит аминокислотную последовательность, отличную от RRNY (SEQ ID NO:101), в позициях, соответствующих аминокислотам 97-100 из SEQ ID NO:18.
Выделенный или рекомбинантный ND может дополнительно определяться по любым из перечисленных выше признакам, которые применимы к полипептидам Nif. Все возможные комбинации перечисленных выше признаков рассматриваются как часть изобретения.
Во взаимосвязанном аспекте настоящее изобретение относится к слитому полипептиду NifD, содержащему митохондриальный нацеливающий пептид (MTP), трансляционно слитой с полипептидом NifD (ND), или его расщепленный продукт, который содержит ND, где слитой полипептид NifD или его расщепленный продукт: (а) устойчив к расщеплению протеазой на сайте в аминокислотной последовательности, соответствующей аминокислотам 97-100 из SEQ ID NO:18; и/или (b) содержит аминокислотную последовательность, отличную от RRNY (SEQ ID NO:101) в позициях, соответствующих аминокислотам 97-100 из SEQ ID NO: 18.
В одном из вариантов осуществления, слитой полипептид NifD содержит олигопептидный линкер и полипептид NifK (NK), которые трансляционно слиты как слитой полипептид NifD-линкер-NifK, где ND содержит С-конец, а NK содержит N-конец, где олигопептидный линкер трансляционно слит с С-концом ND и N-концом NK.
В другом аспекте настоящее изобретение относится к расщепленному продукту слитого полипептида NifD, согласно изобретению, где расщепленный продукт содержит ND, олигопептидный линкер и NK, где олигопептидный линкер трансляционно слит с С-концом. В одном из вариантов осуществления, слитой полипептид NifD или его расщепленный продукт, по меньшей мере, частично растворим в митохондриях растительной клетки, когда слитой полипептид NifD продуцируется в растительной клетке.
В одном из вариантов осуществления, слитой полипептид NifD представляет собой слитой полипептид AnfD, NK представляет собой полипептид AnfK, а слитой полипептид NifD-линкер-NifK представляет собой слитой полипептид AnfD-линкер-AnfK.
В другом аспекте настоящее изобретение относится к слитому полипептиду NifK, содержащему митохондриальный нацеливающий пептид (MTP), трансляционно слитой с полипептидом NifK (NK), где слитой полипептид NifK или его расщепленный продукт, по меньшей мере, частично растворим в митохондриях растительной клетки, когда слитой полипептид NifK или его расщепленный продукт продуцируется в растительной клетке.
В другом аспекте настоящее изобретение относится, согласно изобретению, к расщепленному продукту слитого полипептида NifK, который содержит NK, причем расщепленный продукт, по меньшей мере, частично растворим в митохондриях растительной клетки, когда расщепленный продукт продуцируется в растительной клетке.
В одном из вариантов осуществления, NK представляет собой полипептид AnfK.
В одном из вариантов осуществления, полипептид NifK имеет С-концевую аминокислотную последовательность, которая совпадает с С-концевой аминокислотной последовательностью полипептида NifK дикого типа. Подходящие полипептидные последовательности NifK дикого типа описаны в этом документе.
В другом аспекте настоящее изобретение относится к белковому комплексу, содержащему (i) расщепленный продукт слитого полипептида NifD, (ii) расщепленный продукт слитого полипептида NifK и (iii) кластер Fe-S, предпочтительно P-кластер.
В одном из вариантов осуществления, белковый комплекс находится в растительной клетке, предпочтительно в митохондриях растительной клетки.
В другом аспекте настоящее изобретение относится, по существу, к очищенному или рекомбинантному полипептиду NifV (NV), который при экспрессии в растительной клетке, по меньшей мере, частично растворим в митохондриях растений.
Во взаимосвязанном аспекте настоящее изобретение относится к выделенному или рекомбинантному полипептиду NifV или слитому полипептиду NifV, содержащему митохондриальный нацеливающий пептид (MTP), трансляционно слитой с полипептидом NifV (NV), или его расщепленным продуктом, содержащим NV, при этом полипептид NifV и/или слитой полипептид NifV и/или его расщепленный продукт, по меньшей мере, частично растворим в растительной клетке, когда продуцируется в растительной клетке, и, предпочтительно, по меньшей мере, частично растворим в митохондриях растительной клетки.
В одном из вариантов осуществления, выделенный или рекомбинантный полипептид NifV, или слитой полипептид NifV, или его расщепленный продукт способен продуцировать гомоцитрат в растительной клетке, предпочтительно в митохондриях растительной клетки.
В другом аспекте настоящее изобретение относится, по существу, к очищенному или рекомбинантному полипептиду NifH (NH), который при экспрессии в растительной клетке, предпочтительно в трансгенном растении, по меньшей мере, частично растворим в митохондриях растения.
В другом аспекте настоящее изобретение относится к слитому полипептиду NifH, содержащему митохондриальный нацеливающий пептид (MTP), трансляционно слитой с полипептидом NifH (NH), или его расщепленным продуктом, который содержит NH, где слитой полипептид NifH и/или его расщепленный продукт, по меньшей мере, частично растворим в митохондриях растительной клетки. В некоторых вариантах осуществления каждого из вышеуказанных аспектов, полипептид NH, по меньшей мере, частично расщеплен в своей последовательности MTP в растительной клетке с продуцированием MPP-расщепленного полипептида Nif, где MPP-расщепленный полипептид NH содержит пептид (пептид scar) длиной от 1 до 45 аминокислот, предпочтительно длиной от 1 до 20 аминокислот, более предпочтительно диной от 1 до 11 аминокислот или от 11 до 20 аминокислот, полученных из последовательности MTP, трансляционно слитых в N-концевой области NH. В некоторых вариантах осуществления, один или несколько пептидов scar, независимо, имеют в длину 2, 3, 4, 5, 6, 7, 8, 9 или 10 аминокислот. В некоторых вариантах осуществления, один или несколько пептидов scar, независимо, имеют в длину 11, 12, 13, 14, 15, 16, 17, 18, 19 или 20 аминокислот или 20-30, 20-40 или 20-50 аминокислот, хотя предпочтительны более короткие последовательности scar.
В одном из вариантов осуществления этих аспектов NH представляет собой полипептид AnfH.
В одном варианте осуществления, слитой полипептид NifH или, предпочтительно, его MPP-расщепленный продукт связан с одним или двумя кластерами Fe-S, предпочтительно с одним или двумя кластерами Fe4-S4.
В другом аспекте предусматривается выделенный или экзогенный полинуклеотид, кодирующий полипептид NifV (NV), где NV при экспрессии в растительной клетке, по меньшей мере, частично растворим в митохондриях растений.
В одном из вариантов осуществления, полипептид NV включает аминокислоты, имеющие последовательность, представленную как любую из SEQ ID NO: 163, 206-209, 211 или 212, как его биологически активный фрагмент или имеет аминокислотную последовательность, которая идентична, по меньшей мере, на 30%, идентична, по меньшей мере, на 40% идентична, по меньшей мере, на 50% идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80% идентична, по меньшей мере, на 90% идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99% тому, что представлено в любой одной или нескольких SEQ ID NO: 163, 206-209, 211 или 212.
В одном из вариантов осуществления, полипептид, согласно изобретению, представляет собой выделенный или рекомбинантный полипептид. В другом варианте осуществления, полипептид, согласно изобретению, например, рекомбинантный полипептид, присутствует в клетке, предпочтительно в растительной клетке.
Подходящие аминокислотные последовательности для полипептидов Nif, в отношении любого из вышеперечисленных аспектов, известны специалистам в отрасли техники, к которой относится данное изобретение, и включают те, которые предоставлены в данном документе.
В одном из вариантов осуществления, полипептид NifH содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99% любой одной или нескольким из следующих последовательностей:
SEQ ID NO:1;
SEQ ID NO:218;
SEQ ID NO:224;
Учетный №. WP_049123239.1;
Учетный №. WP_048638817.1;
Учетный №. WP_013029017.1;
Учетный №. WP_013010353.1;
Учетный №. WP_014258951.1;
Учетный №. WP_011744626.1;
Учетный №. WP_013718497.1;
Учетный №. WP_009565928.1;
Учетный №. WP_013099472.1;
Учетный №. WP_007781874.1;
Учетный №. WP_012703362;
Учетный №. WP_153472986;
Учетный №. WP_015854293;
Учетный №. WP_123927773;
Учетный №. WP_073538802; и
Учетный №. RCV6483.
В одном из вариантов осуществления, полипептид NifH содержит один или несколько мотивов аминокислотной последовательности из SEQ ID NO:225-231.
В одном из вариантов осуществления, полипептид NifH содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99%, или идентична последовательности представленной в SEQ ID NO:1.
В одном из вариантов осуществления, полипептид NifH содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99%, или идентична последовательности, представленной в SEQ ID NO:218.
В одном из вариантов осуществления, полипептид NifD содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99% любой одной или нескольким из следующих последовательностей:
SEQ ID NO:2;
SEQ ID NO:18;
SEQ ID NO:148;
SEQ ID NO:149;
SEQ ID NO:150;
SEQ ID NO:151;
SEQ ID NO:152;
SEQ ID NO:153;
SEQ ID NO:216;
Учетный №. WP_044347161.1;
Учетный №. WP_047370273.1;
Учетный №. WP_038902190.1;
Учетный №. WP_024872642.1;
Учетный №. WP_024078601.1;
Учетный №. WP_013298320.1;
Учетный №. WP_010877172.1;
Учетный №. WP_014258953.1;
Учетный №. WP_066665786.1;
Учетный №. WP_015773055.1;
Учетный №. WP_016867598.1;
Учетный №. WP_009512873.1;
Учетный №. WP_012703361;
Учетный №. WP_075356167;
Учетный №. WP_038590013;
Учетный №. WP_ 023922817;
Учетный №. WP_011021232; и
Учетный №. OAV73823.
В одном из вариантов осуществления, полипептид NifD содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99% или идентична последовательности, представленной в SEQ ID NO:2.
В одном из вариантов осуществления, полипептид NifD содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99%, или идентична последовательности представленной в SEQ ID NO:216.
В одном из вариантов осуществления, полипептид NifK содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99% любой одной или нескольким из следующих последовательностей:
SEQ ID NO:3;
SEQ ID NO:217;
Учетный №. WP_049080161.1;
Учетный №. WP_044347163.1;
Учетный №. SBM87811.1;
Учетный №. WP_047370272.1;
Учетный №. WP_014333919.1;
Учетный №. WP_012728880.1;
Учетный №. WP_011912506.1;
Учетный №. WP_065303473.1;
Учетный №. WP_018989051.1;
Учетный №. prf||2106319A;
Учетный №. WP_011021239.1;
Учетный №. WP_012703359;
Учетный №. WP_144571040;
Учетный №. WP_077859050;
Учетный №. WP_122630336; и
Учетный №. WP_088520366.
В одном из вариантов осуществления, полипептид NifK содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99%, или идентична последовательности, представленной в SEQ ID NO:3.
В одном из вариантов осуществления, полипептид NifK содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99%, или идентична последовательности, представленной в SEQ ID NO:217.
В одном из вариантов осуществления, полипептид NifB содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99% любой одной или нескольким из следующих последовательностей:
SEQ ID NO:4;
Учетный №. WP_041145602.1;
Учетный №. WP_043953592.1;
Учетный №. WP_040003311.1;
Учетный №. WP_011094468.1;
Учетный №. WP_048638849.1;
Учетный №. WP_011813098.1;
Учетный №. WP_048108879.1;
Учетный №. WP_050355163.1;
Учетный №. WP_015850328.1; и
Учетный №. P10930.
В одном из вариантов осуществления, полипептид NifB содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99%, или идентична последовательности представленной в SEQ ID NO:4.
В одном из вариантов осуществления, полипептид NifE содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99% любой одной или нескольким из следующих последовательностей:
SEQ ID NO:5;
Учетный №. WP_049114606.1;
Учетный №. SBM87755.1;
Учетный №. WP_012764127.1;
Учетный №. WP_012728883.1;
Учетный №. WP_003297989.1;
Учетный №. WP_012698965.1;
Учетный №. WP_013190624.1;
Учетный №. WP_025698318.1;
Учетный №. WP_013460149.1;
Учетный №. AIS31022.1;
Учетный №. WP_018701501.1; и
Учетный №. WP_048514099.1.
В одном из вариантов осуществления, полипептид NifE содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99%, или идентична последовательности, представленной в SEQ ID NO:5.
В одном из вариантов осуществления, полипептид NifF содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99% любой одной или нескольким из следующих последовательностей:
SEQ ID NO:6;
Учетный №. WP_004122417.1;
Учетный №. WP_040968713.1;
Учетный №. WP_035885760.1;
Учетный №. WP_039999438.1;
Учетный №. WP_048638838.1;
Учетный №. WP_064006977.1;
Учетный №. WP_012698862.1;
Учетный №. WP_010933399.1;
Учетный №. WP_002949173.1; и
Учетный №. WP_039801725.1.
В одном из вариантов осуществления, полипептид NifF содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99%, или идентична последовательности, представленной в SEQ ID NO:6.
В одном из вариантов осуществления, полипептид AnfG содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99% любой одной или нескольким из следующих последовательностей:
SEQ ID NO:219;
Учетный №. WP_012703360;
Учетный №. WP_144571041;
Учетный №. HBE76208;
Учетный №. WP_144349445;
Учетный №. WP_112317428; и
Учетный №. WP_048515315.
В одном из вариантов осуществления, полипептид AnfG содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99%, или идентична последовательности представленной в SEQ ID NO:219.
В одном из вариантов осуществления, полипептид NifJ содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99% любой одной или нескольким из следующих последовательностей:
SEQ ID NO:7;
Учетный №. WP_024360006.1;
Учетный №. WP_044347157.1;
Учетный №. WP_050533844.1;
Учетный №. WP_064566543.1;
Учетный №. WP_057084649.1;
Учетный №. WP_014683040.1;
Учетный №. WP_013149847.1;
Учетный №. WP_053341220.1;
Учетный №. WP_014454638.1; и
Учетный №. CSA83023.1.
В одном из вариантов осуществления, полипептид NifJ содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99%, или идентична последовательности, представленной в SEQ ID NO:7.
В одном из вариантов осуществления, полипептид NifM содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99% любой одной или нескольким из следующих последовательностей:
SEQ ID NO:8;
Учетный №. WP_064342940.1;
Учетный №. WP_004122413.1;
Учетный №. WP_044347181.1;
Учетный №. WP_064566543.1;
Учетный №. WP_063105800.1;
Учетный №. WP_035885759.1;
Учетный №. WP_011094472.1;
Учетный №. WP_048638837.1;
Учетный №. CAA75544.1;
Учетный №. WP_051692859.1; и
Учетный №. WP_018415157.1.
В одном из вариантов осуществления, полипептид NifM содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99%, или идентична последовательности, представленной в SEQ ID NO:8.
В одном из вариантов осуществления, полипептид NifN содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99% любой одной или нескольким из следующих последовательностей:
SEQ ID NO:9;
Учетный №. WP_064391778.1;
Учетный №. WP_047370268.1;
Учетный №. WP_014683026.1;
Учетный №. WP_048638830.1;
Учетный №. WP_027147663.1;
Учетный №. WP_015195966.1;
Учетный №. WP_023593609.1;
Учетный №. WP_025677480.1; и
Учетный №. WP_018306265.1.
В одном из вариантов осуществления, полипептид NifN содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99%, или идентична последовательности представленной в SEQ ID NO:9.
В одном из вариантов осуществления, полипептид NifQ содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99% любой одной или нескольким из следующих последовательностей:
SEQ ID NO:10;
Учетный №. WP_064391765.1;
Учетный №. CTQ06350.1;
Учетный №. WP_047370257.1;
Учетный №. WP_043878077.1;
Учетный №. WP_008878174.1;
Учетный №. WP_011501504.1;
Учетный №. WP_027196569.1;
Учетный №. GAU06296.1; и
Учетный №. WP_063239464.1.
В одном из вариантов осуществления, полипептид NifQ содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99%, или идентична последовательности, представленной в SEQ ID NO:10.
В одном из вариантов осуществления, полипептид NifS содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99% любой одной или нескольким из следующих последовательностей:
SEQ ID NO:11;
SEQ ID NO:19;
Учетный №. WP_004138780.1;
Учетный №. WP_045858151.1;
Учетный №. WP_047370265.1;
Учетный №. WP_014333911.1;
Учетный №. WP_055731597.1;
Учетный №. WP_014239770.1;
Учетный №. WP_054691765.1;
Учетный №. WP_021802294.1;
Учетный №. WP_026894054.1; и
Учетный №. WP_061575621.1.
В одном из вариантов осуществления, полипептид NifS содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99%, или идентична последовательности, представленной в SEQ ID NO:11.
В одном из вариантов осуществления, полипептид NifS содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99%, или идентична последовательности, представленной в SEQ ID NO:19.
В одном из вариантов осуществления, полипептид NifU содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99% любой одной или нескольким из следующих последовательностей:
SEQ ID NO:12;
Учетный №. WP_049136164.1;
WP_050887862.1;
WP_057084657.1;
WP_048638833.1;
WP_012728889.1;
WP_055731596.1;
WP_028587630.1;
WP_044417303.1;
WP_001051984.1; и
KIM05011.1.
В одном из вариантов осуществления, полипептид NifU содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99%, или идентична последовательности, представленной в SEQ ID NO:12.
В одном из вариантов осуществления, полипептид NifV содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99% любой одной или нескольким из следующих последовательностей:
SEQ ID NO:13;
SEQ ID NO:163;
SEQ ID NO:164;
SEQ ID NO:206;
SEQ ID NO:207;
SEQ ID NO:208;
SEQ ID NO:209;
SEQ ID NO:210;
SEQ ID NO:211;
SEQ ID NO:212;
SEQ ID NO:213;
SEQ ID NO:214;
SEQ ID NO:215;
Учетный №. WP_049083341.1;
Учетный №. WP_045858154.1;
Учетный №. WP_047370264.1;
Учетный №. WP_038912041.1;
Учетный №. WP_048638835.1;
Учетный №. WP_011712856.1;
Учетный №. WP_037528703.1;
Учетный №. OAA29062.1; и
Учетный №. EKQ56006.1.
В одном из вариантов осуществления, полипептид NifV содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99%, или идентична последовательности, представленной в SEQ ID NO:13.
В одном из вариантов осуществления, полипептид NifX содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99% любой одной или нескольким из следующих последовательностей:
SEQ ID NO:14;
Учетный №. WP_049070199.1;
Учетный №. WP_064342937.1;
Учетный №. WP_044347173.1;
Учетный №. WP_044612922.1;
Учетный №. WP_043953583.1;
Учетный №. WP_039999416.1;
Учетный №. WP_047608097.1;
Учетный №. WP_039800848.1;
Учетный №. WP_062149047.1; и
Учетный №. WP_020165972.1.
В одном из вариантов осуществления, полипептид NifX содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99%, или идентична последовательности, представленной в SEQ ID NO:14.
В одном из вариантов осуществления, полипептид NifY содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99% любой одной или нескольким из следующих последовательностей:
SEQ ID NO:15;
Учетный №. WP_049089500.1;
Учетный №. WP_064342935.1;
Учетный №. WP_044524054.1;
Учетный №. WP_049010739.1;
Учетный №. WP_047370270.1;
Учетный №. WP_039999411.1;
Учетный №. WP_037382461.1;
Учетный №. WP_014683024.1;
Учетный №. AEX25784.1; и
Учетный №. WP_012698835.1.
В одном из вариантов осуществления, полипептид NifY содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99%, или идентична последовательности, представленной в SEQ ID NO:15.
В одном из вариантов осуществления, полипептид NifZ содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99% любой одной или нескольким из следующих последовательностей:
SEQ ID NO:16;
Учетный №. WP_057173223.1;
Учетный №. WP_064342939.1;
Учетный №. WP_043875005.1;
Учетный №. WP_043953588.1;
Учетный №. WP_065368553.1;
Учетный №. WP_062627625.1;
Учетный №. WP_011491838.1;
Учетный №. WP_014029050.1; и
Учетный №. WP_015665422.1.
В одном из вариантов осуществления, полипептид NifZ содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99%, или идентична последовательности, представленной в SEQ ID NO:16.
В одном из вариантов осуществления, полипептид NifW содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99% любой одной или нескольким из следующих последовательностей:
SEQ ID NO:17;
Учетный №. WP_064342938.1;
Учетный №. WP_049080155.1;
Учетный №. WP_095103586.1;
Учетный №. WP_065877373.1;
Учетный №. WP_095699971.1;
Учетный №. WP_012764136.1;
Учетный №. WP_053085547.1;
Учетный №. WP_077299824.1;
Учетный №. OGI40729;
Учетный №. ACO76430.1; и
Учетный №. BBA37427.1.
В одном из вариантов осуществления, полипептид NifW содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99%, или идентична последовательности, представленной в SEQ ID NO:17.
В одном из вариантов осуществления, полипептид ферредоксин содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99% любой одной или нескольким из следующих последовательностей:
SEQ ID NO:232;
Учетный №. WP_012703542;
Учетный №. WP_065835964.1;
Учетный №. WP_069124666.1;
Учетный №. WP_101942980;
Учетный №. WP_049076934.1;
Учетный №. WP_072048756.1;
Учетный №. WP_130674512.1; и
Учетный №. WP_103805005.1.
В одном из вариантов осуществления, полипептид ферредоксин содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99%, или идентична последовательности, представленной в SEQ ID NO:232.
Подходящие аминокислотные последовательности для MTP, в отношении любого из вышеперечисленных аспектов, известны специалистам в отрасли техники, к которой относится данное изобретение, и включают те, которые предоставлены в данном документе. В одном из вариантов осуществления, MTP содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99% любой одной или нескольким из следующих последовательностей:
SEQ ID NO:36;
SEQ ID NO:21;
аминокислоты 1-77 из SEQ ID NO:20;
SEQ ID NO:28;
SEQ ID NO:29;
SEQ ID NO:30;
SEQ ID NO:31;
SEQ ID NO:32;
SEQ ID NO:33;
SEQ ID NO:34;
SEQ ID NO:35;
SEQ ID NO:37; и
SEQ ID NO:38.
В одном из вариантов осуществления, MTP содержит аминокислоты, имеющие последовательность, которая идентична, по меньшей мере, на 30% идентична, по меньшей мере, на 40%, идентична, по меньшей мере, на 50%, идентична, по меньшей мере, на 60%, идентична, по меньшей мере, на 70%, идентична, по меньшей мере, на 80%, идентична, по меньшей мере, на 90%, идентична, по меньшей мере, на 95%, идентична, по меньшей мере, на 97% или идентична, по меньшей мере, на 99%, или идентична последовательности, представленной в SEQ ID NO:36.
В другом аспекте настоящее изобретение касается полинуклеотида, кодирующего любой один или несколько полипептидов.
В одном из вариантов осуществления, кодирующая белок область полинуклеотида была кодон-модифицирована для экспрессии в растительной клетке, по сравнению с соответствующей кодирующей белок областью встречающегося в природе полинуклеотида в бактерии. В одном из вариантов осуществления, большая часть или даже все кодирующие белок области были кодон-оптимизированы для экспрессии в растительной клетке, предпочтительно растительной клетке согласно изобретению.
В дополнительном варианте осуществления, каждый экзогенный полинуклеотид содержит промотор, функционально связанный с полинуклеотидом, и/или элементами трансляционной регуляции, функционально связанными с полинуклеотидом.
В другом варианте осуществления, промотор обеспечивает экспрессию одного или нескольких полинуклеотидов в корнях, листьях и/или стебле растения, предпочтительно промотор обеспечивает экспрессию одного или нескольких полинуклеотидов в одном или нескольких, или во всех корнях, листьях или стебле растения, относительно семян растения.
В другом варианте осуществления, один или несколько или все полинуклеотиды присутствующие в растительной клетке или бактериальной клетке, предпочтительно интегрированы в ядерный геном растительной клетки, например, в виде сцепленной последовательности DNA, интегрированной в хлоропластный геном или, предпочтительно, в ядерный геном растительной клетки. Растительная клетка может содержать несколько копий сцепленной последовательности DNA, интегрированной в ядерный геном, например, в виде нескольких T-DNA.
В одном из вариантов осуществления, каждый полинуклеотид или каждая последовательность в нем, кодирующая полипептид, функционально связана с промотором и, необязательно, с последовательностью терминации транскрипции.
В дополнительном или другом варианте осуществления, промотор обеспечивает экспрессию одного или нескольких полинуклеотидов в корнях, листьях и/или стебле растения, предпочтительно, один или нескольких полинуклеотидов экспрессируются в одном или нескольких, или во всех корнях, листьях или стебле растения, относительно семян растения.
В дополнительном аспекте предусматривается химерный вектор, содержащий или кодирующий полинуклеотид, согласно изобретению.
В другом аспекте настоящее изобретение касается вектора, содержащего полинуклеотид согласно изобретению.
В одном из вариантов осуществления, вектор содержит полинуклеотиды, которые кодируют, по меньшей мере, 3, по меньшей мере, 4 или, по меньшей мере, 5 слитых полипептидов Nif.
В другом аспекте настоящее изобретение касается вектора, содержащего полинуклеотиды, которые кодируют, по меньшей мере, 3, по меньшей мере, 4 или, по меньшей мере, 5 слитых полипептидов Nif, определенных в любом из вышеуказанных аспектов изобретения.
В одном из вариантов осуществления, вектор содержит полинуклеотиды, которые кодируют:
а) слитой полипептид NifD и слитой полипептид NifK или слитой полипептид NifD-линкер-NifK; и
b) слитой полипептид NifH и слитой полипептид NifV; и,
c) необязательно, слитой полипептид AnfG и/или слитой полипептид ферредоксин.
В одном из вариантов осуществления, вектор содержит полинуклеотиды, которые кодируют
а) слитые полипептиды NifF, NifJ, NifU и NifB и, необязательно, слитой полипептид NifS; и/или
b) слитые полипептиды NifW, NifX, NifY и NifZ.
В дополнительном аспекте настоящее изобретение относится к клетке, содержащей один или несколько полипептидов согласно изобретению, один или несколько экзогенных полинуклеотидов согласно изобретению и/или вектор согласно изобретению.
В одном из вариантов осуществления, клетка представляет собой растительную клетку или бактериальную клетку.
В дополнительном варианте осуществления, растительная клетка представляет собой односеменодольную растительную клетку, например, клетку злакового растения, например, клетку пшеницы, клетку риса, клетку кукурузы, клетку тритикале, клетку овса или клетку ячменя, предпочтительно клетку пшеницы или двусемядольную растительную клетку. Растительная клетка может быть дополнительно охарактеризована полипептидами или полинуклеотидами, определяемыми любым из перечисленных выше признаков. Все возможные комбинации перечисленных выше признаков рассматриваются как часть изобретения в контексте растительной клетки и других аспектов изобретения.
В дополнительном аспекте настоящее изобретение относится к трансгенному растению или его трансгенной части, предпочтительно семени, содержащих один или несколько полипептидов, согласно изобретению, один или несколько экзогенных полинуклеотидов, согласно изобретению, и/или вектор согласно изобретению.
В одном из вариантов осуществления, трансгенное растение представляет собой односеменодольное растение, например, злаковое растение, например, пшеницу, рис, кукурузу, тритикале, овес или ячмень, предпочтительно, пшеницу или двусемядольное растение. Растение или его часть может быть дополнительно охарактеризована полипептидами или полинуклеотидами, определяемыми любым из перечисленных выше признаков. Все возможные комбинации перечисленных выше признаков рассматриваются как часть изобретения в контексте растения или его части, а также в других аспектах изобретения.
В дополнительном аспекте настоящее изобретение относится к способу продуцирования полипептида согласно изобретению, способу, включающему экспрессию полинуклеотида в клетке, согласно изобретению.
В дополнительном аспекте настоящее изобретение относится к способу продуцирования клетки, согласно изобретению, способу, включающему стадию введения в клетку одного или нескольких полинуклеотидов, согласно изобретению, и/или вектора согласно изобретению.
В другом аспекте настоящее изобретение относится к способу продуцирования гомоцитрата в растительной клетке, способу включающему экспрессию рекомбинантного полипептида NifV или слитого полипептида NifV, согласно изобретению, в растительной клетке, в котором рекомбинантный полипептид NifV или слитой полипептид NifV, и/или его расщепленный продукт продуцирует гомоцитрат в растительной клетке.
В одном из вариантов осуществления, способ дополнительно включает введение в растительную клетку полинуклеотида, кодирующего рекомбинантный полипептид NifV или слитой полипептид NifV.
В другом аспекте настоящее изобретение относится к применению полипептида NifV, согласно изобретению, для продуцирования гомоцитрата в растительной клетке.
В другом аспекте настоящее изобретение относится к способу увеличения количества слитого полипептида NifD, NifK или NifD-линкер-NifK в растительной клетке, при этом способ включает экспрессию одного или нескольких или всех слитых полипептидов NifW, NifX, NifY и NifZ в растительной клетке, где каждый слитой полипептид Nif независимо содержит митохондриальный нацеливающий пептид (MTP), причем количество слитого полипептида NifD, NifK или NifD-линкер-NifK в растительной клетке увеличено по сравнению с соответствующей растительной клеткой, не экспрессирующей один или несколько или все слитые полипептиды NifW, NifX, NifY и NifZ.
В одном из вариантов осуществления, способ дополнительно включает:
i) введение в растительную клетку одного или нескольких полинуклеотидов, кодирующих слитой полипептид NifD, NifK или NifD-линкер-NifK; и
ii) введение в растительную клетку одного или нескольких полинуклеотидов, кодирующих один или несколько или все слитые полипептиды NifW, NifX, NifY и NifZ.
В другом аспекте настоящее изобретение относится к способу увеличения количества полипептида NifY в растительной клетке, при этом способ включает экспрессию одного или нескольких или всех слитых полипептидов NifW, NifX и NifZ в растительной клетке, где каждый слитой полипептид Nif независимо содержит митохондриальный нацеливающий пептид (MTP), где количество полипептида NifY в растительной клетке увеличено по сравнению с соответствующей растительной клеткой, не экспрессирующей один или несколько или все слитые полипептиды NifW, NifX и NifZ.
В одном из вариантов осуществления, способ дополнительно включает:
i) введение в растительную клетку полинуклеотида, кодирующего слитой полипептид NifY; и
ii) введение в растительную клетку одного или нескольких полинуклеотидов, кодирующих один или несколько или все слитые полипептиды NifW, NifX и NifZ.
В другом аспекте настоящее изобретение относится к использованию одного или нескольких полинуклеотидов, кодирующих один или несколько или все слитые полипептиды NifW, NifX и NifZ, для увеличения количества полипептида NifY в растительной клетке.
В другом аспекте настоящее изобретение относится к использованию полинуклеотида, согласно изобретению, и/или вектора, согласно изобретению, для продуцирования трансгенной растительной клетки.
В другом аспекте настоящее изобретение относится к способу продуцирования трансгенного растения, при этом способ включает стадии:
i) введение в растительную клетку одного или нескольких полинуклеотидов, согласно изобретению, и/или одного или нескольких векторов, согласно изобретению,
ii) регенерации, из клетки стадии i), трансгенного растения, согласно изобретению, и
iii) продуцирование, необязательно, трансгенных семян и/или растений-потомков из трансгенного растения, регенерированного на стадии ii).
В дополнительном аспекте настоящее изобретение относится к способу продуцирования трансгенных семян, включающему:
i) сбор семян трансгенного растения, согласно изобретению, и/или
ii) сбор семян от одного или нескольких трансгенных растений-потомков, полученных способом согласно изобретению.
В дополнительном аспекте настоящее изобретение относится к способу продуцирования растения, которое интегрировало в свой геном полинуклеотид, согласно изобретению, способу включающему стадии:
i) скрещивания двух родительских растений, где, по меньшей мере, одно растение содержит полинуклеотид,
ii) скрининг одного или нескольких растений-потомков, полученных от скрещивания, на наличие или отсутствие полинуклеотида, и
iii) отбор растения-потомка, содержащего полинуклеотид,
тем самым продуцируя растение.
В дополнительном или другом варианте осуществления, по меньшей мере, одно из родительских растений представляет собой тетраплоидное или гексаплоидное растение пшеницы.
В дополнительном или другом варианте осуществления, стадия ii) включает анализ на полинуклеотид образца, содержащего DNA одного или нескольких растений-потомков.
В дополнительном или другом варианте осуществления, стадия iii) включает:
i) отбор растения-потомка, гомозиготного по отношению полинуклеотиду, и/или
ii) анализ растения или одного или нескольких его растений- потомков на наличие и/или экспрессию полинуклеотида или на измененный фенотип, как определено выше.
В одном или дополнительном варианте осуществления, способ дополнительно включает:
iv) возвратное скрещивание растений-потомков, после скрещивания на стадии i), с растением того же генотипа, что и первое родительское растение, но не имеющее полинуклеотида, достаточное количество раз для продуцирования растения с большей частью генотипа первого родителя, но содержащего полинуклеотид, и
v) отбор растения-потомка, которое содержит полинуклеотид и/или имеет измененный фенотип, как определено выше.
В следующем или другом варианте осуществления, способ дополнительно включает стадию анализа растения или растения потомка, по меньшей мере, на один другой генетический маркер.
В дополнительном аспекте настоящее изобретение относится к растению, продуцированному с использованием способа согласно изобретению.
В дополнительном аспекте настоящее изобретение относится к применению полинуклеотида, согласно изобретению, и/или вектора, согласно изобретению, для продуцирования рекомбинантной клетки и/или трансгенного растения.
В одном из вариантов осуществления, трансгенное растение имеет измененный фенотип, как определено выше, по сравнению с соответствующим растением, лишенным экзогенного полинуклеотида и/или вектора.
В дополнительном аспекте настоящее изобретение относится к способу идентификации растения, содержащего полинуклеотид согласно изобретению, способу, включающему стадии:
i) получения образца нуклеиновой кислоты из растения, и
ii) скрининга образца на наличие или отсутствие полинуклеотида.
В одном из вариантов осуществления, наличие полинуклеотида указывает на то, что растение имеет измененный фенотип, как определено выше, по сравнению с соответствующим растением, лишенным экзогенного полинуклеотида.
В дополнительном или другом варианте осуществления, способ идентифицирует растение согласно изобретению.
В дополнительном или другом варианте осуществления, способ дополнительно включает продуцирование растения из семян перед этапом i).
В другом аспекте настоящее изобретение относится к части трансгенного растения, содержащую растительную клетку согласно изобретению или полученную из трансгенного растения согласно изобретению.
В одном из вариантов осуществления, часть растения представляет собой семя, которое содержит полинуклеотид согласно изобретению.
В другом аспекте настоящее изобретение относится к способу производства муки, муки крупного помола, крахмала, масла, муки из семян или другого продукта, полученного из семян, способу включающему:
а) получение семян согласно изобретению и/или
b) экстрагирование муки, муки крупного помола, крахмала, масла или другого продукта или производство муки из семян.
В дополнительном аспекте настоящее изобретение относится к продукту, продуцированному из трансгенного растения согласно изобретению и/или части растения согласно изобретению, включающему полипептид согласно изобретению и/или полинуклеотид согласно изобретению.
В одном из вариантов осуществления, часть растения представляет собой семя.
В дополнительном или другом варианте осуществления, продукт представляет собой пищевой ингредиент или ингредиент напитка, или пищевой продукт, или напиток. Предпочтительно чтобы, i) пищевой ингредиент или продукт выбирался из группы, состоящей из: муки, крахмала, масла, дрожжевого или пресного хлеба, макаронных изделий, лапши, кормов для животных, хлопьев для завтрака, закусок, тортов, солода, выпечки и пищевых продуктов, содержащих соусы на основе муки, или ii) напиток, представляющий собой сок, пиво или солод. Способы производства таких продуктов хорошо известны специалистам в отрасли техники, к которой относится данное изобретение.
В альтернативном варианте осуществления, продукт представляет собой не пищевой продукт. Примеры не пищевых продуктов включают, не ограничиваясь, пленки, покрытия, клеи, строительные материалы и упаковочные материалы. Способы производства таких продуктов хорошо известны специалистам в отрасли техники, к которой относится данное изобретение.
В дополнительном аспекте настоящее изобретение относится к способу приготовления пищевого продукта, способу включающему: смешивание с другим пищевым ингредиентом семян согласно изобретению или муки, муки крупного помола, крахмала, масла или другого продукта из семян.
В дополнительном аспекте настоящее изобретение относится к способу получения солода, включающему стадию проращивания семян согласно изобретению.
В дополнительном аспекте настоящее изобретение относится к использованию растения или его части в соответствии с изобретением в качестве корма для животных или для производства корма для потребления животными или пищи для потребления человеком.
В дополнительном аспекте настоящее изобретение относится к композиции, содержащей полипептид согласно изобретению, полинуклеотид согласно изобретению, вектор согласно изобретению или клетку согласно изобретению и один или несколько приемлемых носителей.
В дополнительном аспекте настоящее изобретение относится к способу реконструкции белкового комплекса нитрогеназы в растительной клетке, способу включающему введение в клетку двух или более полинуклеотидов согласно изобретению, двух или более конструкций нуклеиновых кислот согласно изобретению и/или вектора согласно изобретению и культивирование растительной клетки в течение времени, достаточного для экспрессии полинуклеотидов или вектора.
Любой вариант осуществления в данном документе следует применять mutatis mutandis к любому другому варианту осуществления, если специально не указано иное. Например, что очевидно для специалиста, примеры полипептидов Nif, описанные выше для одного аспекта изобретения, в равной степени применимы к любому из других аспектов изобретения.
Настоящее изобретение не ограничивается в объеме описанными здесь конкретными вариантами осуществления, которые предназначены только для иллюстрации. Функционально эквивалентные продукты, композиции и способы явно входят в объем изобретения, как описано здесь.
В данном описании, если специально не указано иное или контекст не требует иного, ссылка на одну стадию, состав вещества, группу стадий или группу составов вещества охватывает одну и несколько (т. е. как минимум одну) из этих стадий, составов вещества, групп стадий или групп составов вещества.
Далее изобретение описывается с помощью следующих неограничивающих примеров и со ссылкой на прилагаемые рисунки.
КРАТКОЕ ОПИСАНИЕ РИСУНКОВ
Рисунок 1. Анализ вестерн-блот с использованием антитела анти-НА для обнаружения отдельных непроцессированных и MPP-процессированных полипептидов pFAγ51::Nif::HA or 6×HIS::Nif::HA после транзиентной экспрессии в листьях Nicotiana benthamiana. C - цитоплазматическая экспрессия (6xHis); M - митохондриально нацеленный.
Рисунок 2. Анализ вестерн-блот белковых экстрактов после введения генетических конструкций MTP: Nif в клетки листьев N. benthamiana. Первая и последняя дорожки на каждой мембране для блоттинга показывают индикативные маркеры молекулярной массы в кДа с использованием чередования полос последовательности нуклеотидов полученных в аппарате Invitrogen Prestained BenchMark ladder. Генетическая конструкция (и), используемая для каждого образца, указывается над каждой дорожкой, а полипептид Nif, включенный в каждый слитой полипептид, указывается под дорожками. Для конструкций SN26-SN32 парные инфильтрирования осуществляли с или без совместного инфильтрирования pRA25, которая кодирует слитой полипептид MTP-FAγ77::NifK (WO2018/141030). Анализ вестерн-блот проводили с помощью антитела HA.
Рисунок 3. Анализ вестерн-блот с использованием антитела анти-НА отдельных полипептидов MTP-FAγ51::Nif::HA (за исключением MTP-FAγ51::HA::NifK) и их MPP-процессированных продуктов, после экспрессии в клетках листа Nicotiana benthamiana. T, суммарный белок; I, нерастворимая фракция; S, растворимая фракция.
Рисунок 4. На верхней панели показана схема генетических конструкций, исследованных на продуцирование продукта вторичного расщепления из слитых полипептидов NifD дикого типа. MTP представлял собой последовательность FAγ51 или L29, а NifD представлял собой последовательность K. oxytoca дикого типа, а HA=эпитоп HA. Внизу показан анализ вестерн-блот белковых экстрактов после введения генетических конструкций в клетки листьев N. benthamiana. Анализ вестерн-блот проводили с помощью антитела HA. Дорожка 1 показывает маркеры молекулярной массы с использованием чередования полос последовательности нуклеотидов, полученных в аппарате Prestained Benchmark ladder. Парные дорожки показывают либо отсутствие (-), либо присутствие (+) конструкции pRA25 NifK. Полоса 1 представляет собой непроцессированный слитой полипептид MTP::NifD, полоса 2 представляет собой MPP-процессированный слитой полипептид, а полоса 3 представляет собой продукт деградации массой ~ 48 кДа.
Рисунок 5. Анализ вестерн-блот белковых экстрактов после введения генетических конструкций MTP:NifD в клетки листьев N. benthamiana. Дорожка 1 показывает маркеры молекулярной массы в кДа с использованием чередования полос последовательности нуклеотидов, полученных в аппарате ThermoFisher Prestained Benchmark ladder. Генетическая конструкция, использованная в каждом образце, указана над каждой дорожкой. pRA24 кодирует полипептид MTP-FAγ::NifD::HA, где кодирующая область NifD была кодон-оптимизирована для Arabidopsis (WO2018/141030). Каждую конструкцию вводили в растительные клетки вместе с pRA25 (MTP-FAγ77::NifK) для усиления накопления слитого полипептида NifD. Анализ вестерн-блот проводили с помощью антитела HA. Стрелка показывает позицию массой ~ 48 кДа полипептида вторичного расщепления из NifD.
Рисунок 6. Анализ вестерн-блот белковых экстрактов после введения генетических конструкций MTP:NifD в клетки листьев N. benthamiana. Дорожка 1 показывает маркеры молекулярной массы в кДа с использованием чередования полос последовательности нуклеотидов, полученных в аппарате ThermoFisher Prestained Benchmark ladder. Генетическая конструкция, использованная в каждом образце, указана над каждой дорожкой. SN64 кодировала полипептид mMTP-CPN60::NifD, в котором аминокислотная последовательность mMTP-CPN60 была изменена в результате замены аминокислот на аланины, что сделало его устойчивым к расщеплению MPP. Конструкция pRA24 кодировала полипептид MTP-FAγ::NifD::HA, где кодирующая область NifD была кодон-оптимизирована для Arabidopsis (WO2018/141030). Анализ вестерн-блот проводили с помощью антитела HA.
Рисунок 7. Выравнивание мутантной аминокислотной последовательности mMTP-FAγ51 (SEQ ID NO:59) в SN66, с немодифицированной последовательностью MTP-FAγ51 (SEQ ID NO:21) в SN10 (SEQ ID NO:122). Участки из 5 и 8 последовательных аминокислотных остатков были заменены аланинами, чтобы инактивировать процессинг MPP.
Рисунок 8. Анализ вестерн-блот белковых экстрактов после введения генетических конструкций MTP:Nif в растительные клетки или дрожжевые клетки, проводили с помощью антитела HA, который показал вторичное расщепление/деградацию NifD в дрожжевых клетках и уменьшение расщепления с аминокислотной заменой Y100Q (SN114, SNY114). Белковые экстракты из клеток листьев N. benthamiana (SN10, SN196, SN114) или из дрожжей (SNY10, SNY196, SNY114) подвергали электрофорезу, как указано в дорожках. Дорожки 1 и 8 показывают маркеры молекулярной массы в кДа с использованием чередования полос последовательности нуклеотидов, полученных в аппарате ThermoFisher Prestained Benchmark ladder. Полоса массой ~ 64 кДа представляет собой непроцессированный слитой полипептид MTP::NifD::HA, полоса массой ~ 58 кДа представляет собой MPP-процессированный слитой полипептид. Стрелка указывает на С-концевой полипептид с массой ~ 48 кДа, продуцируемый вторичным расщеплением.
Рисунок 9. Анализ вестерн-блот белковых экстрактов из клеток листьев N. benthamiana после введения генетических конструкций, кодирующих варианты аминокислотной замены MTP::NifD::HA, каждый вместе с SN46 (MTP-Su9::NifK). Дорожка 12 показывает маркеры молекулярной массы в кДа с использованием чередования полос последовательности нуклеотидов, полученных в аппарате ThermoFisher Prestained Benchmark ladder. Наиболее интенсивной полосой массой ~ 58 кДа на дорожках 5-11 была MTP-FAγ51::NifD, процессированная MPP. Дорожки 2 и 3 показывают полипептид массой 48 кДа, продуцированный вторичным расщеплением. Обратите внимание на отсутствие полипептида массой 48 кДа в дорожках 5-11.
Рисунок 10. Выравнивание аминокислотной последовательности из области полипептидов NifD дикого типа, соответствующей аминокислотам 49-108 K. oxytoca NifD (SEQ ID NO:18). Репрезентативную последовательность выбирали из каждого кластера, который содержал, по меньшей мере, 10 элементов в сети сходства последовательностей. Количество элементов в каждом кластере последовательностей NifD показано в скобках. Полностью консервативные аминокислоты показаны над выравниванием.
Рисунок 11. Локализация предполагаемого сайта вторичного расщепления показана в кристаллической структуре полипептида NifD из K. oxytoca (PDB:1QGU). FeMo-кофактор показан в виде сфер справа. NifK-Ser515, NifK-Asp517, С-конец и структуры в верхнем левом углу взяты из полипептида NifK. Arg97, Arg98, Asn99, Tyr100, Tyr101, Thr102 и структуры справа внизу, кроме FeMo-ко, взяты из NifD. Пунктирными линиями показаны возможные водородные связи между гидроксилом Tyr100 и Ser515, Asp517 и Arg98.
Рисунок 12. Анализ вестерн-блот показывает процессинг митохондрий слитых полипептидов NifD от шести различных бактерий. Три конструкции на соседних дорожках были проанализированы для каждой последовательности NifD: кодирующие слитой полипептид mMTP-FAγ51::NifD::HA, который не расщеплялся MPP в каноническом сайте расщепления MPP (дорожки, отмеченные A); MTP-FAγ51::NifD::HA, который был нацелен на митохондрии (дорожки, обозначенные M); и 6xHis::NifD::HA, который, как ожидалось, должен был локализоваться в цитоплазме (дорожки, обозначенные C) и соответствовать размеру процессированного в MPP.
Рисунок 13. Схематические карты генетических конструкций, кодирующих слитые полипептиды NifD::linker(HA)::NifK представлены не в масштабе. mMTP-FAγ относится к мутантному MTP, имеющему замены аланина для предотвращения расщепления MPP. Y100Q относится к наличию аминокислотной замены в последовательности NifD.
Рисунок 14. Растворимость полипептидов NifD-линкер(НА)-NifK после экспрессии в N. benthamiana. Белки из образцов инфильтрированных листьев выделяли как «общий» белок или фракционировали на нерастворимые и растворимые фракции, как описано в Примере 1. Маркер белка, показанный с использованием чередования полос последовательности нуклеотидов, полученных в аппарате ThermoFisher Prestained Benchmark ladder, использовался в мембранах для блоттинга для «общих» и «нерастворимых» образцов, а чередования полос последовательности нуклеотидов, полученных в аппарате Invitrogen PageRuler использовалась в мембране для блоттинга для «растворимых» образцов.
Рисунок 15. Схема слитого полипептида с метаксином, кодируемого геном В SN197, и его локализация во внешней мембране митохондрий с большей частью полипептида от N-конца в цитоплазму. Эта конструкция использовала последовательность метаксина N. benthamiana.
Рисунок 16. Анализ вестерн-блот показал, что очистка митохондриально нацеленного MTP-FAγ51::NifU::TS из SN166 привела к очистке процессированной формы полипептида NifU. Верхняя панель: проводили с помощью антитела анти-Strep. Нижняя панель: Гель кумасси синий.
Рисунок 17. Анализ вестерн-блот показал, что очистка митохондриально нацеленного scar9::GG::NifU::TS привела к совместной очистке scar9::GG::NifS::HA. Образцы со стадий (i) - (v) в процессе очистки первого эксперимента по очистке подвергали электрофорезу SDS-PAGE и анализу вестерн-блот с использованием либо антитела анти-Strep для обнаружения полипептида NifU, либо антитела анти-HA для обнаружения полипептида NifS. Две полосы для NifS соответствуют непроцессированным и процессированным формам. Присутствие в элюате процессированной формы NifS показало, что произошла совместная очистка.
Рисунок 18. Анализ вестерн-блот очистки NifU из N. benthamiana в третьем эксперименте по очистке, показывающий, что NifS очищается совместно с NifU. Панель A) Схема конструкций, которые были инфильтрированы в N. benthamiana (не в масштабе). B) Очистка - анализ вестерн-блот. P=осадок, S=надосадочная жидкость, FT=протекание и E=элюат. Все образцы загружали в двух экземплярах и подвергали иммуно-детекции с использованием либо антител-strep (α-strep), либо антител-НА (α-НА). C) Окрашивание элюата гелем кумасси показывает полосу большой интенсивности для NifU и полосу небольшой интенсивности для NifS.
Рисунок 19. Анализ вестерн-блот показал, что очистка митохондриально нацеленного MTP-FAγ51::NifS::TS привела к совместной очистке scar9::GG::NifU::HA. Образцы со стадий (ii) - (v) подвергали электрофорезу SDS-PAGE и анализу вестерн-блот с использованием либо антитела анти-Strep для обнаружения полипептида NifS, либо антитела анти-HA для обнаружения полипептида NifU. Две полосы для NifS соответствуют непроцессированным и процессированным формам. Присутствие в элюате процессированной формы NifU показало, что произошла совместная очистка.
Рисунок 20. Программа CаlustalW выравнивания первых 300 аминокислотных остатков из выбранных NifV/HCS-подобных аминокислотных последовательностей в этом исследовании равно как и N. benthamiana P72026 (SEQ ID NO:221) и P20586 (SEQ ID NO:222) трансляция, K. oxytoca NifV (SEQ ID NO:13), Lotus japonicus FEN1 (SEQ ID NO:215), и Mycobacterium tuberculosis α-изопропилмалатсинтазы (MtLeuA, SEQ ID NO:223). Другие последовательности HCS взяты из Thermoanaerobacter brockii (TbHCS; SEQ ID NO:206), Thermincola potens (TpHCS; SEQ ID NO:207), Saccharomyces cerevisiae (ScHCS; SEQ ID NO:208), Nodularia spumigena(NsHCS; SEQ ID NO:209), Methanosarcina acetivorans (MaHCS; SEQ ID NO:210), Chlorobaculum tepidum (CtHCS; SEQ ID NO:211) и Methanocaldococcus infernus (MiHCS1, SEQ ID NO:212; MiHCS2, SEQ ID NO:213; MiHCS, SEQ ID NO:214). Консервативные остатки в активном сайте LeuA обозначены *. Четыре аминокислотных остатка в позициях R81, D82, H291, H293 содержат Zn2+, а два аминокислотных остатка E224, T260 равно как и Zn2+ в его позиции образуют субстрат-связывающий карман MtLeuA (Koon et al., 2004).
Рисунок 21. Анализ вестерн-блот, использующий антитело анти-НА общих, нерастворимых и растворимых фракций NifV/HCS-подобных слитых полипептидов (MTP-FAγ51::HA::NifV/HCS) после экспрессии в листьях N. benthamiana. Т - общий белок; I, нерастворимая (осадочная) фракция общего белка; S, растворимая (надосадочная) фракция общего белка. m, митохондриально нацеленный полипептид; c, цитоплазмо нацеленный полипептид.
Рисунок 22. Анализ вестерн-блот, использующий антитело анти-HA общих, нерастворимых и растворимых фракций цитоплазматически локализованных NifV/HCS-подобных слитых полипептидов (HA::NifV/HCS) после экспрессии в листьях N. benthamiana, используемых в качестве компараторов для соответствующих митохондриально- локализованных слитых полипептидов. Т - общий белок; I, нерастворимая (осадочная) фракция общего белка; S, растворимая (надосадочная) фракция общего белка; c, цитоплазмо нацеленный полипептид; m, митохондриально нацеленный полипептид.
Рисунок 23. Площадь пика целевого иона гомоцитрата после вычитания базовой линии (шкала Log10).
Рисунок 24. Анализ вестерн-блот растворимости слитых полипептидов NifH в системе для транзиентной экспрессии листьев N. benthamiana с использованием антитела анти-Strep для обнаружения полипептидов, имеющих эпитоп TwinStrep. Все генетические конструкции NifH были совместно инфильтрированы с SN44, кодирующей слитой полипептид NifM из K. oxytoca. Образцы белка приготавливали в аэробных условиях.
Рисунок 25. Анализ вестерн-блот, показывающий результаты очистки слитого полипептида NifH, кодированного SL6, в стабильно трансформированном табаке. Ген NifH кодировал слитой полипептид MTP-CoxIV::TwinStrep::KoNifH::HA. Образцы объемом 5 мкл со стадий процесса очистки подвергали анализу вестерн-блот и проводили с помощью антител, распознающих эпитопы Strep или HA. Над дорожками указаны образцы из общей, нерастворимой и растворимой фракций. Неокрашенные стрелки указывают на непроцессированный полипептид NifH, а черные стрелки указывают на процессированную форму.
Рисунок 26. Анализ вестерн-блот экспрессии и процессинга слитых полипептидов Anf после транзиентного введения генетических конструкций в листья N. benthamiana. Мембрана для блоттинга имела наборы из трех смежных дорожек для (слева направо) слитых полипептидов AnfD, AnfK, AnfH и AnfG. Каждый набор включал исследуемый слитой полипептид MTP-FAγ51::HA::Anf и два контрольных полипептида HA::Anf и mFAγ51::HA::Anf в качестве маркеров молекулярной массы. L, последовательность маркеров молекулярной массы (кДа).
Рисунок 27. Анализ вестерн-блот, показывающий экспрессию и процессинг всех четырех слитых полипептидов AnfD, AnfK, AnfH и AnfG при экспрессии из мультигенных конструкций в листьях N. benthamiana. A. Анализ вестерн-блот митохондриально нацеленнных слитых полипептидов AnfD, AnfK, AnfG и AnfH, экспрессированных из SL26, и непроцессированных полипептидов из SL31, обнаруженных в экстрактах общего белка при анализе транзиентной экспрессии листьев. B. Анализ вестерн-блот белков, полученных в результате экспрессии митохондриально нацеленных слитых полипептидов AnfD, AnfK, AnfG и AnfH из SL26 и непроцессированных слитых полипептидов из SL31. C. Анализ вестерн-блот, показывающий экспрессию и процессинг слитых полипептидов из мультигенных конструкций SL26, SL27 и SL28, конструкции с одним геном SL29 и смеси (Mix) из четырех конструкций с одним геном SN161, SN129, SN130 и SN131. При наличии AnfK показывает верхнюю непроцессированную полосу и нижнюю процессированную полосу.
Рисунок 28. Анализ вестерн-блот, показывающий растворимость отдельных полипептидов Anf, экспрессированных из векторов с одним геном в клетках листьев N. benthamiana, при локализации в цитоплазме или митохондриях. На верхних панелях показаны растворимые фракции слитых полипептидов AnfD, AnfK, AnfH и AnfG, а на нижних панелях показаны нерастворимые фракции слитых полипептидов AnfD, AnfK, AnfH и AnfG. С - цитоплазматическая локализация; М - митохондриальная локализация; A, аланин, замещенный mMTP-FAγ51. Черные стрелки указывают позиции МРР-расщепленных белков, неокрашенные стрелки указывают на непроцессированные полипептиды. См. Таблицу 20 для прогнозируемых молекулярных масс каждого Anf в непроцессированных и MPP-процессированных полипептидах.
Рисунок 29. Модель гомологии комплекса AnfDKHG для Fe-нитрогеназы, основанная на аминокислотных последовательностях Anf A. vinelandii с линкером, соединяющим полипептиды AnfD и AnfK. Начальные координаты прежде 20 нс моделирования. Спрогнозированная структура полипептида AnfD::Linker::AnfK с использованием линкера из 16 аминокислот была связана в комплекс с димерами AnfH и AnfG. Димер AnfH обозначен как AnfHH.
Рисунок 30. Анализ вестерн-блот экстрактов общего белка из листьев N. benthamiana, инфильтрированных генетическими конструкциями для экспрессии полипептидов AnfD и AnfK, либо слитых, либо раздельных. Мембрану для блоттинга анализировали антителом анти-НА. Экспрессию слитых полипептидов AnfD-линкер-AnfK из SN272-SN275 сравнивали с экспрессией отдельных генов в векторах SL26 и SL28. Конструкции SN161 и SN129 обеспечивали контроль экспрессии по отдельности для AnfD и AnfK, соответственно.
Рисунок 31. Анализ вестерн-блот (А) растворимых и (В) нерастворимых фракций белков из листьев N. benthamiana, инфильтрированных с генетическими конструкциями для экспрессии генов AnfD и AnfK. Каждая из конструкций SN272-SN275 кодировала слитые полипептиды AnfD-линкер-AnfK, тогда как SL26 и SL28 экспрессировали отдельные полипептиды.
Рисунок 32. Анализ вестерн-блот полипептидов, продуцированных из SL42 в листьях N. benthamiana,включая общие (Т), нерастворимые (I) и растворимые (S) фракции, с использованием антител анти-НА (панель A) или антител анти-Strep (панель B) для обнаружения. Черные стрелки указывают положения полос процессированного полипептида после митохондриального расщепления MPP, белые стрелки указывают полосы для непроцессированных полипептидов. Панель B показывает процессированный полипептид NifB, анализ проводили с помощью антитела анти-Strep.
Рисунок 33. Анализ вестерн-блот полипептидов, продуцированных из SL43 в листьях N. benthamiana,включая общие (Т), нерастворимые (I) и растворимые (S) фракции, с использованием антител анти-НА (панель A) или антител анти-Strep (панель B) для обнаружения. Черные стрелки указывают положения полос процессированного полипептида после митохондриального расщепления MPP, белые стрелки указывают полосы для непроцессированных полипептидов. Панель B показывает процессированный полипептид AnfK, анализ проводили с помощью антитела анти-Strep.
Рисунок 34. Анализ вестерн-блот полипептидов, продуцированных из SL42 и SL43 и введенных вместе в листья N. benthamiana, включая общие (Т), нерастворимые (I) и растворимые (S) фракции, с использованием антител анти-НА (панель A) или антител анти-Strep (панель B) для обнаружения. . Цифры сбоку от панелей A) и B) отображают молекулярные массы (кДа) маркеров на первой дорожке. Черные стрелки указывают положения полос процессированного полипептида после митохондриального расщепления MPP, белые стрелки указывают полосы для непроцессированных полипептидов.
Рисунок 35. Анализ вестерн-блот полипептидов, продуцированных из SL48 в листьях N. benthamiana, включая общие (Т), нерастворимые (I) и растворимые (S) фракции, с использованием антител анти-НА (панель A) или антител анти-Strep (панель B) для обнаружения. Цифры сбоку от панелей A) и B) отображают молекулярные массы (кДа) маркеров на первой дорожке. Черные стрелки указывают положения полос процессированного полипептида после митохондриального расщепления MPP, белые стрелки указывают полосы для непроцессированных полипептидов. Панель B показывает процессированный полипептид NifB, анализ проводили с помощью антитела анти-Strep.
Рисунок 36. Анализ вестерн-блот полипептидов, продуцированных из SL49 в листьях N. benthamiana,включая общие (Т), нерастворимые (I) и растворимые (S) фракции, с использованием антител анти-НА (панель A) или антител анти-Strep (панель B) для обнаружения. Черные стрелки указывают положения полос процессированного полипептида после митохондриального расщепления MPP, белые стрелки указывают полосы для непроцессированных полипептидов. Панель B показывает процессированный полипептид AnfK, анализ проводили с помощью антитела анти-Strep.
Рисунок 37. Анализ вестерн-блот полипептидов, продуцированных из SL48 и SL49 введенных вместе в листья N. benthamiana, включая общие (Т), нерастворимые (I) и растворимые (S) фракции, с использованием антител анти-НА (панель A) или антител анти-Strep (панель B) для обнаружения. Черные стрелки указывают положения полос процессированного полипептида после митохондриального расщепления MPP, белые стрелки указывают полосы для непроцессированных полипептидов.
Рисунок 38. Анализ вестерн-блот полипептидов, продуцированных из SN292, SN291, SN299 и SN300 в листьях N. benthamiana, включая общие, панель A), нерастворимые, панель B) и растворимые, панель C), фракции, с использованием анти-HA для обнаружения. Цифры сбоку от отображают молекулярные массы (кДа) маркеров на первой дорожке. Черные стрелки указывают положения полос процессированного полипептида после митохондриального расщепления, белые стрелки указывают полосы для непроцесированных полипептидов, * указывает потенциальный димер белка FdxN.
Рисунок 39. Анализ вестерн-блот полипептидов, продуцированных из SN192, SL50 и SL54, введенных по отдельности, а также SL50 и SL54 введенных вместе в листья N. benthamiana, включая Общие (панель A), Растворимые (панель B) и Нерастворимые (панель C) фракции с использованием анти-HA для обнаружения. Черные стрелки указывают положения полос процессированного полипептида после митохондриального расщепления, белые стрелки указывают полосы для непроцессированных полипептидов.
Рисунок 40. Анализ вестерн-блот полипептидов, продуцированных из SL50 в листьях N. benthamiana, включая общие, панель A), нерастворимые, панель B) и растворимые, панель C), фракции, с использованием анти-HA для обнаружения. Черные стрелки указывают положения полос процессированного полипептида после митохондриального расщепления, белые стрелки указывают полосы для непроцессированных полипептидов.
Рисунок 41. Анализ вестерн-блот полипептидов, продуцированных из SL50 и SL49 в листьях N. benthamiana, включая общие, панель A), нерастворимые, панель B) и растворимые, панель C), фракции, с использованием анти-HA для обнаружения. Черные стрелки указывают положения полос процессированного полипептида после митохондриального расщепления, белые стрелки указывают полосы для непроцессированных полипептидов.
Рисунок 42. Анализ вестерн-блот полипептидов, продуцированных из SL47 и SL55, по отдельности или в комбинации, в листьях N. benthamiana с использованием анти-НА для обнаружения. На первой дорожке показаны маркеры молекулярной массы (кДа). Черные стрелки указывают положения полос процессированного полипептида после митохондриального расщепления MPP, белые стрелки указывают полосы для непроцессированных полипептидов.
КЛЮЧ К СПИСКУ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
SEQ ID NO:1 Аминокислотная последовательность полипептида NifH из K. oxytoca, 293aa.
SEQ ID NO:2 Аминокислотная последовательность полипептида дикого типа NifD из K. oxytoca, согласно Учетного №. X13303.1; 483aa (Последовательность Temme представляет собой SEQ ID NO:18).
SEQ ID NO:3 Аминокислотная последовательность полипептида of NifK из K. oxytoca, согласно работы Temme et al. (2012); 520aa.
SEQ ID NO:4 Аминокислотная последовательность полипептида NifB из K. oxytoca, 468aa.
SEQ ID NO:5 Аминокислотная последовательность полипептида NifE из K. oxytoca, 457aa.
SEQ ID NO:6 Аминокислотная последовательность полипептида NifF из K. oxytoca, 176 aa; NCBI Учетный №. X03214.
SEQ ID NO:7 Аминокислотная последовательность полипептида NifJ из K. oxytoca, 1171 aa; NCBI Учетный №. 43862;Cannon et al., 1988 Nucleic Acids Res. 16:11379).
SEQ ID NO:8 Аминокислотная последовательность полипептида NifM из K. oxytoca, 266 aa; NCBI Учетный №. X05887; Paul и Merrick (1987).
SEQ ID NO:9 Аминокислотная последовательность полипептида NifN из K. oxytoca, NCBI Учетный №. P08738; 461aa; (Arnold et al., 1988). Эта последовательность идентична последовательности K. michiganensis Учетный №. WP_064371582 и на 85% идентична последовательности, обозначенной как K. oxytoca NifN, Учетный №. WP_061153953.
SEQ ID NO:10 Аминокислотная последовательность полипептида NifQ из Klebsiella. NCBI Учетный №. WP_004138772. Эта последовательность на 95% идентична другой последовательности K. Oxytoca,, обозначенной как NifQ, Учетный №. AAA25108.1.
SEQ ID NO:11 Аминокислотная последовательность полипептида NifS из K. oxytoca, 400aa.
SEQ ID NO:12 Аминокислотная последовательность полипептида NifU из K. oxytoca, 274aa. NCBI Учетный №. P05343.2 (Arnold et al., 1988). Эта последовательность идентична последовательности под Учетным №. WP_004138782, а 272/273 также идентичны другой последовательности K. Oxytoca,, Учетный №. AAA25155.
SEQ ID NO:13 Аминокислотная последовательность полипептида NifV из K. oxytoca, 381aa. NCBI Учетный №. CAA31119.1 (Arnold et al., 1988).
SEQ ID NO:14 Аминокислотная последовательность полипептида NifX из K. oxytoca, 156aa (Учетный №. P09136).
SEQ ID NO:15 Аминокислотная последовательность полипептида NifY из K. Oxytoca,, 220aa; NCBI Учетный №. CAA31670 (Arnold et al., 1988).
SEQ ID NO:16 Аминокислотная последовательность полипептида NifZ из K. Oxytoca,148aa; NCBI Учетный №. P0A3U2 (Arnold et al., 1988).
SEQ ID NO:17. Аминокислотная последовательность полипептида NifW из K. oxytoca.
SEQ ID NO:18. Аминокислотная последовательность NifD дикого типа из K. Oxytoca,, в соответствии с работой Temme et al. (2012).
SEQ ID NO:19. Аминокислотная последовательность NifS дикого типа из K. Oxytoca,, в соответствии с работой Temme et al. (2012).
SEQ ID NO:20. Аминокислотная последовательность N-концевого продолжения, содержащая MTP-FAγ77 (аминокислоты 1-77) и триплетные аминокислоты GAP (78-80). Расщепление MPP происходит между аминокислотными остатками 42 и 43.
SEQ ID NO:21. Аминокислотная последовательность полипептида MTP-FAγ51 с дополнительным N-концом Met и C-концом GG. Расщепление MPP происходит между аминокислотными остатками 43 и 44.
SEQ ID NO:22. Аминокислотная последовательность полипептида FAγ-scar9.
SEQ ID NO:23. Аминокислотная последовательность слитого полипептида MTP-FAγ77::NifH::HA, кодированного pRA10. Аминокислоты 1-77 соответствуют MTP-FAγ77, аминокислоты 78-80 являются GAP, аминокислоты 81-372 соответствуют аминокислотам NifH K. oxytoca (SEQ ID NO:1 без инициатора Met), а аминокислоты 373-389 содержат эпитоп НА.
SEQ ID NO:24. Аминокислотная последовательность слитого полипептида MTP-FAγ51::NifH::HA, кодированного pRA34. Аминокислоты 1-51 соответствуют MTP-FAγ51, аминокислоты 52-54 являются GAP, аминокислоты 55-346 соответствуют NifH K. oxytoca (SEQ ID NO:1 без инициатора Met), а аминокислоты 347-363 содержат эпитоп НА.
SEQ ID NO:25. Аминокислотная последовательность слитого полипептида MTP-FAγ51::NifH::HA, кодированного SN18. Аминокислоты 1-54 соответствуют MTP-FAγ51 с GG, аминокислоты 55-347 соответствуют NifH K. oxytoca (SEQ ID NO:1), а аминокислоты 348-358 содержат эпитоп HA.
SEQ ID NO:26. Аминокислотная последовательность слитого полипептида MTP-FAγ51::HA::NifH, кодированного SN29. Аминокислоты 1-53 соответствуют MTP-FAγ51 с GG, аминокислоты 54-64 содержат эпитоп НА, аминокислоты 65-357 соответствуют NifH K. oxytoca (SEQ ID NO:1), а аминокислоты 358-371 представляли собой C-концевое продолжение.
SEQ ID NO:27. Последовательность 6xHis использовали вместо последовательности MTP, с N-концевым Met и C-концевым GG.
SEQ ID NO:28. Аминокислотная последовательность CPN60 MTP.
SEQ ID NO:29. Аминокислотная последовательность CPN60/без линкера GG MTP.
SEQ ID NO:30. Аминокислотная последовательность супероксид-дисмутазы (SOD) MTP.
SEQ ID NO:31. Аминокислотная последовательность удвоенной супероксид-дисмутазы (2SOD) MTP.
SEQ ID NO:32. Аминокислотная последовательность модифицированной супероксид-дисмутазы, (SODmod) MTP.
SEQ ID NO:33. Аминокислотная последовательность удвоенной модифицированной супероксид-дисмутазы, (2SODmod) MTP.
SEQ ID NO:34. Аминокислотная последовательность L29 MTP (At1G07830).
SEQ ID NO:35. Аминокислотная последовательность 9 субъединицы F0 ATPase (SU9) MTP из Neurospora crassa.
SEQ ID NO:36. Аминокислотная последовательность гамма-субъединицы gATPase (FAγ51) MTP без дополнительного N-концевого Met (SEQ ID NO:21 имеет дополнительный N-концевой Met). Расщепление MPP происходит между аминокислотными остатками 42 и 43.
SEQ ID NO:37. Аминокислотная последовательность CoxIV twin strep (ABM97483) MTP.
SEQ ID NO:38. Аминокислотная последовательность CoxIV 10xHis (ABM97483) MTP.
SEQ ID NO:39. Аминокислотная последовательность предсказанной scar для супероксид-дисмутазы (SOD) MTP с GG и для удвоенной супероксид-дисмутазы (2SOD) MTP с GG.
SEQ ID NO:40. Аминокислотная последовательность предсказанной scar для L29 MTP с GG.
SEQ ID NO:41. Аминокислотная последовательность предсказанной scar для 9 субъединицы F0 ATPase (SU9) MTP из Neurospora crassa с GG.
SEQ ID NO:42. Аминокислотная последовательность предсказанной scar для гамма-субъединицы gATPase (FAγ51 ) MTP с GG.
SEQ ID NO:43. Аминокислотная последовательность предсказанной scar для CoxIV twin strep MTP с GG.
SEQ ID NO:44. Аминокислотная последовательность предсказанной scar для CoxIV 10xHis MTP с GG.
SEQ ID NO:45. Олигонуклеотидный праймер MIT_V2.1_SbfInifH_FW2.
SEQ ID NO:46. Олигонуклеотидный праймер MIT_V2.1_SbfInifJ_RV2.
SEQ ID NO:47. Олигонуклеотидный праймер MIT_V2.1_SbfInifB_FW.
SEQ ID NO:48. Олигонуклеотидный праймер MIT_V2.1_SbfIori_RV.
SEQ ID NO:49. Аминокислотная последовательность mscar9 из MTP-FAγ51 с замещением N-концевого остатка Ile на Met для инициирования трансляции.
SEQ ID NO:50. Триптический пептид.
SEQ ID NO:51. Аминокислотная последовательность MTP-FAγ9 scar без N-концевой Met и с C-концевой Met.
SEQ ID NOs:52-54. Олигонуклеотидные праймеры.
SEQ ID NO:55. Триптический пептид.
SEQ ID NO:56. Триптический пептид.
SEQ ID NO:57. Аминокислотная последовательность слитого полипептида (pRA25) MTP-FAγ77::NifK без какого-либо С-концевого продолжения. Аминокислоты 1-77 соответствуют MTP-FAγ77, аминокислоты 78-80 представляют собой GAP, а аминокислоты 81-599 соответствуют NifK K. oxytoca без инициатора Met.
SEQ ID NO:58. Аминокислотная последовательность последних четырех аминокислотных остатков на С-конце полипептида NifK из K. oxytoca.
SEQ ID NO:59. Аминокислотная последовательность мутантного полипептида MTP-FAγ51, который не расщепляется MPP.
SEQ ID NOs:60-107. Пептидные последовательности.
SEQ ID NOs:108-113. Олигонуклеотидные праймеры.
SEQ ID NO:114. Аминокислотная последовательность участка из 11 остатков линкерной области целлобиогидролазы II Hypocrea jecorina (Учетный № AAG39980.1).
SEQ ID NO:115. Аминокислотная последовательность из 9-остатков эпитопа HA.
SEQ ID NO:116. Аминокислотная последовательность линкера для слитого пептида NifD::линкер::NifK. Линкер имеет в длину 30 остатков и имеет SEQ ID NO:114 с концевым аргинином, замененным на аланин, затем 9-остатков эпитопа HA (SEQ ID NO:115), за которым следует другая копия SEQ ID NO: 114 с аргинином, замененным на аланин.
SEQ ID NO:117. Олигонуклеотидный праймер.
SEQ ID NO:118. Олигонуклеотидный праймер.
SEQ ID NO:119. Пептидная последовательность scar.
SEQ ID NO:120. Пептидная последовательность scar.
SEQ ID NO:121. Аминокислотная последовательность слитого полипептида с метаксином, кодированная конструкцией SN197. Эпитоп TwinStrep соответствует аминокислотам 1-31, mTurquoise соответствует аминокислотам 32-273, сайт расщепления TEV соответствует аминокислотам 274-282, а последовательность метаксина соответствует аминокислотам 283-603.
SEQ ID NO:122 Аминокислотная последовательность слитого полипептида MTP-FAγ51::NifD::HA, кодированная SN10. Аминокислоты 1-54 соответствуют MTP-FAγ51 с GG на его С-конце, аминокислоты 55-536 соответствуют NifD K. oxytoca (SEQ ID NO:18) с его инициатором Met, а аминокислоты 537-547 содержат эпитоп HA.
SEQ ID NO:123. Аминокислотная последовательность слитого полипептида MTP-FAγ51::NifM::HA, кодированная SN30. Аминокислоты 1-54 соответствуют MTP-FAγ51 с GG на его С-конце, аминокислоты 55-320 соответствуют NifM K. oxytoca (SEQ ID NO:8) с его инициатором Met, а аминокислоты 321-331 содержат эпитоп HA.
SEQ ID NO:124. Аминокислотная последовательность слитого полипептида MTP-FAγ51::NifS::HA, кодированная SN31. Аминокислоты 1-54 соответствуют MTP-FAγ51 с GG на его С-конце, аминокислоты 55-454 соответствуют K. oxytoca NifS (SEQ ID NO:19) с его инициатором Met, согласно работе Temme et al. (2012), а аминокислоты 455-465 содержат эпитоп HA.
SEQ ID NO:125. Аминокислотная последовательность слитого полипептида MTP-FAγ51::NifU::HA, кодированная SN32. Аминокислоты 1-54 соответствуют MTP-FAγ51 с GG на его С-конце, аминокислоты 55-328 соответствуют K. oxytoca NifU (SEQ ID NO:12) с его инициатором Met, а аминокислоты 329-339 содержат эпитоп HA.
SEQ ID NO: 126. Аминокислотная последовательность слитого полипептида MTP-FAγ51::NifE::HA, кодированная SN38. Аминокислоты 1-54 соответствуют MTP-FAγ51 с GG на его С-конце, аминокислоты 55-511 соответствуют K. oxytoca NifE с его инициатором Met, согласно работе Temme et al. (2012), а аминокислоты 512-522 включают эпитоп HA.
SEQ ID NO:127. Аминокислотная последовательность слитого полипептида MTP-FAγ51::NifN::HA, кодированная SN39. Аминокислоты 1-54 соответствуют MTP-FAγ51 с GG на его С-конце, аминокислоты 55-515 соответствуют K. oxytoca NifN (SEQ ID NO:9) с его инициатором Met, а аминокислоты 516-526 содержат эпитоп HA.
SEQ ID NO:128. Аминокислотная последовательность слитого полипептида MTP-CoxIV-Twin-Strep::NifH::HA, кодированная SN42. Аминокислоты 1-61 соответствуют MTP-CoxIV-Twin-Strep с GG на его С-конце, аминокислоты 62-354 соответствуют K. oxytoca аминокислотам NifH (SEQ ID NO:1) с его инициатором Met, а аминокислоты 355-365 содержат эпитоп HA.
SEQ ID NO: 129. Аминокислотная последовательность слитого полипептида MTP-Su9::NifK, кодированная SN46. Аминокислоты 1-70 соответствуют MTP-Su9 с GG на его С-конце, аминокислоты 71-590 соответствуют K. oxytoca NifK (SEQ ID NO:3) с его инициатором Met.
SEQ ID NO:130. Аминокислотная последовательность слитого полипептида MTP-L29::NifV::HA, кодированная SN51. Аминокислоты 1-34 соответствуют MTP-L29 с GG на его С-конце, аминокислоты 35-415 соответствуют K. oxytoca NifV (SEQ ID NO:13) с его инициатором Met, а аминокислоты 416-426 содержат эпитоп HA.
SEQ ID NO:131. Аминокислотная последовательность слитого полипептида MTP-FAγ51::NifD::линкер(HA)::NifK, кодированная SN68. Аминокислоты 1-54 соответствуют MTP-FAγ51 с GG на его C-конце, аминокислоты 55-536 соответствуют аминокислотам NifD K. oxytoca дикого типа (SEQ ID NO:18 без N-концевого Met), аминокислоты 537-566 соответствуют линкеру, содержащему эпитоп НА, а аминокислоты 567-1085 соответствуют NifK (SEQ ID NO: 3) без его N-концевого Met и с его С-концом дикого типа.
SEQ ID NO:132. Аминокислотная последовательность слитого полипептида MTP-FAγ51::HA::NifD::HA, кодированная SN75. Аминокислоты 1-53 соответствуют MTP-FAγ51 с GG на его С-конце, аминокислоты 54-64 соответствуют первому эпитопу НА, аминокислоты 65-546 соответствуют аминокислотам NifD K. oxytoca дикого типа (SEQ ID NO:18), а аминокислоты 547-557 содержат эпитоп НА.
SEQ ID NO:133. Аминокислотная последовательность слитого полипептида MTP-FAγ51::NifD::HA, кодированная SN99. Аминокислоты 1-54 соответствуют MTP-FAγ51 с GG на его C-конце, аминокислоты 55-536 соответствуют NifD K. oxytoca, содержащему мутации аланина по типу замены в аминокислотах 148-152, а аминокислоты 537-547 содержат эпитоп НА.
SEQ ID NO:134. Аминокислотная последовательность слитого полипептида MTP-FAγ51::NifD::HA, кодированная SN100. Аминокислоты 1-54 соответствуют MTP-FAγ51 с GG на его C-конце, аминокислоты 55-536 соответствуют аминокислотам NifD K. oxytoca, содержащим мутации аланина по типу замены в аминокислотах 153-157, а аминокислоты 537-547 содержат эпитоп НА.
SEQ ID NO:135. Аминокислотная последовательность слитого полипептида MTP-Su9::NifW, кодированная SN104. Аминокислоты 1-70 соответствуют MTP-Su9 с GG на его С-конце, аминокислоты 71-158 соответствуют K. oxytoca NifW (SEQ ID NO:17) с его инициатором Met, а аминокислоты 159-167 содержат эпитоп HA.
SEQ ID NO:136. Аминокислотная последовательность слитого полипептида MTP-FAγ51::NifD::HA, кодированная SN114. Аминокислоты 1-54 соответствуют MTP-FAγ51 с GG на его C-конце, аминокислоты 55-536 соответствуют NifD K. oxytoca, содержащему мутацию Y100Q по типу замены в аминокислоте 154, а аминокислоты 537-547 содержат эпитоп НА.
SEQ ID NO:137. Аминокислотная последовательность слитого полипептида MTP-FAγ51::NifF::HA, кодированная SN138. Аминокислоты 1-54 соответствуют MTP-FAγ51 с GG, аминокислоты 55-230 соответствуют NifF K. oxytoca (SEQ ID NO:6), а аминокислоты 231-241 содержат эпитоп HA.
SEQ ID NO:138. Аминокислотная последовательность слитого полипептида MTP-FAγ51::NifJ::HA, кодированная SN139. Аминокислоты 1-54 соответствуют MTP-FAγ51 с GG, аминокислоты 55-1225 соответствуют NifJ K. oxytoca (SEQ ID NO:7), а аминокислоты 1226-1236 содержат эпитоп HA.
SEQ ID NO:139. Аминокислотная последовательность слитого полипептида MTP-FAγ51::HA::NifK, кодированная SN140. Аминокислоты 1-53 соответствуют MTP-FAγ51 с GG, аминокислоты 54-64 содержат эпитоп НА, а аминокислоты 65-584 соответствуют NifK K. oxytoca (SEQ ID NO:3) с С-концом дикого типа.
SEQ ID NO:140. Аминокислотная последовательность слитого полипептида MTP-FAγ51::NifQ::HA, кодированная SN141. Аминокислоты 1-54 соответствуют MTP-FAγ51 с GG, аминокислоты 55-221 соответствуют NifQ K. oxytoca (SEQ ID NO:10), а аминокислоты 222-232 содержат эпитоп HA.
SEQ ID NO:141. Аминокислотная последовательность слитого полипептида MTP-FAγ51::NifV::HA, кодированная SN142. Аминокислоты 1-54 соответствуют MTP-FAγ51 с GG, аминокислоты 55-435 соответствуют NifV K. oxytoca (SEQ ID NO:13), а аминокислоты 436-446 содержат эпитоп HA.
SEQ ID NO:142. Аминокислотная последовательность слитого полипептида MTP-FAγ51::NifW::HA, кодированная SN143. Аминокислоты 1-54 соответствуют MTP-FAγ51 с GG, аминокислоты 55-140 соответствуют NifW K. oxytoca (SEQ ID NO:17), а аминокислоты 141-151 содержат эпитоп HA.
SEQ ID NO:143. Аминокислотная последовательность слитого полипептида MTP-FAγ51::NifX::HA, кодированная SN144. Аминокислоты 1-54 соответствуют MTP-FAγ51 с GG, аминокислоты 55-210 соответствуют NifX K. oxytoca (SEQ ID NO:14), а аминокислоты 211-221 содержат эпитоп HA.
SEQ ID NO:144. Аминокислотная последовательность слитого полипептида MTP-FAγ51::NifY::HA, кодированная SN145. Аминокислоты 1-54 соответствуют MTP-FAγ51 с GG, аминокислоты 55-274 соответствуют NifY K. oxytoca, согласно работы Temme et al. (2012), а аминокислоты 275-285 содержат эпитоп HA.
SEQ ID NO:145. Аминокислотная последовательность слитого полипептида MTP-FAγ51::NifZ::HA, кодированная SN146. Аминокислоты 1-54 соответствуют MTP-FAγ51 с GG, аминокислоты 55-202 соответствуют NifZ K. oxytoca (SEQ ID NO:16), а аминокислоты 203-213 содержат эпитоп HA.
SEQ ID NO:146. Аминокислотная последовательность слитого полипептида MTP-FAγ51::NifD(Y100Q)::linker(HA)::NifK, кодированная SN159. Аминокислоты 1-54 соответствуют MTP-FAγ51 с GG на его C-конце, аминокислоты 55-536 соответствуют NifD K. oxytoca с замещением Y100Q, аминокислоты 537-566 соответствуют линкеру, содержащему эпитоп НА, а аминокислоты 567-1085 соответствуют NifK (SEQ ID NO:3) без его N-концевого Met и с его С-концом дикого типа.
SEQ ID NO:147. Аминокислотная последовательность слитого полипептида MTP-FAγ51::NifB::HA, кодированная SN192. Аминокислоты 1-54 соответствуют MTP-FAγ51 с GG, аминокислоты 55-522 соответствуют NifB K. oxytoca, согласно работы Temme et al. (2012), а аминокислоты 523-533 содержат эпитоп HA.
SEQ ID NO:148. Аминокислотная последовательность полипептида NifD дикого типа Azospirillum brasilense, UniProt A0A060DN91; 479aa.
SEQ ID NO:149. Аминокислотная последовательность полипептида NifD дикого типа Azotobacter vinelandii, UniProt C1DGZ7; 492aa.
SEQ ID NO:150. Аминокислотная последовательность полипептида NifD дикого типа Sinorhizobium fredii, 504aa.
SEQ ID NO:151. Аминокислотная последовательность полипептида NifD дикого типа Chlorobium tepidum, UniProt Q8KC89; 543aa.
SEQ ID NO:152. Аминокислотная последовательность полипептида NifD дикого типа Desulfovibrio vulgaris, UniProt B8DR77; 544aa.
SEQ ID NO:153. Аминокислотная последовательность полипептида NifD дикого типа Desulfotomaculum ferrireducens, 539aa.
SEQ ID NO:154. Пептидная последовательность, где X представляет собой любую аминокислоту, отличную от Tyr.
SEQ ID NO:155. Последовательность триптического пептида из NifM.
SEQ ID NO:156. Последовательность триптического пептида из NifM.
SEQ ID NO:157. Последовательность триптического пептида из CAT.
SEQ ID NO:158. Последовательность триптического пептида из CAT.
SEQ ID NO:159. Последовательность триптического пептида из CAT.
SEQ ID NO:160. Аминокислотная последовательность слитого полипептида MTP-FAγ51::NifU::TwinStrep, кодированная SN166. Аминокислоты 1-54 представляют собой последовательность MTP-FAγ51 с дополнительным началом трансляции метионина и C-концевым GG, аминокислоты 55-328 представляют собой последовательность NifU, а аминокислоты 329-358 представляют собой последовательность, включающую мотив Twinstrep.
SEQ ID NO:161. Аминокислотная последовательность слитого полипептида MTP-FAγ51::NifS::TwinStrep, кодированная SN231. Аминокислоты 1-54 представляют собой последовательность MTP-FAγ51 с дополнительным началом трансляции метионина и C-концевым GG, аминокислоты 55-454 представляют собой последовательность NifS а аминокислоты 455-484 представляют собой последовательность, включающую мотив Twinstrep.
SEQ ID NO:162. Последовательность триптического пептида из scar9.
SEQ ID NO:163. Аминокислотная последовательность полипептида NifV из A. vinelandii (AvNifV; Учетный №. WP_012698855).
SEQ ID NO:164. Аминокислотная последовательность вариантной последовательности KoNifV (Учетный №. WP_004138778).
SEQ ID NO:165. N-концевое ScHCS продолжение (последовательность scar).
SEQ ID NO:166. N-концевое AvNifV продолжение (последовательность scar).
SEQ ID NO:167. Аминокислотная последовательность полипептида MTP-FAγ51::HA::KoNifM, кодированная SN43. Аминокислоты 1-53 соответствуют последовательности MTP-FAγ51, включая GG на его C-конце, аминокислоты 54-64 соответствуют эпитопу HA, включая GG на его C-конце, а аминокислоты 65-330 соответствуют последовательности NifM из K. oxytoca.
SEQ ID NO:168. Аминокислотная последовательность полипептида MTP-CoxIV::TwinStrep::NifH, кодированная SN178. Аминокислоты 1-31 соответствуют последовательности MTP-CoxIV, аминокислоты 32-61 соответствуют последовательности TwinStrep, включая GG на ее С-конце, а аминокислоты 62-354 соответствуют последовательности NifH из Azospirillum brasilense (Учетный №. WP_014239786).
SEQ ID NO:169. Аминокислотная последовательность полипептида MTP-CoxIV::TwinStrep::NifH, кодированная SN179. Аминокислоты 1-31 соответствуют последовательности MTP-CoxIV, аминокислоты 32-61 соответствуют последовательности TwinStrep, включая GG на ее С-конце, а аминокислоты 62-356 соответствуют последовательности NifH из Mastigocladus laminosus (Учетный №. WP_016865872).
SEQ ID NO:170. Аминокислотная последовательность полипептида MTP-CoxIV::TwinStrep::NifH, кодированная SN180. Аминокислоты 1-31 соответствуют последовательности MTP-CoxIV, аминокислоты 32-61 соответствуют последовательности TwinStrep, включая GG на ее С-конце, а аминокислоты 62-348 соответствуют последовательности NifH из Frankia casurinae (Учетный №. WP_0011438842).
SEQ ID NO:171. Аминокислотная последовательность полипептида MTP-CoxIV::TwinStrep::NifH, кодированная SN181. Аминокислоты 1-31 соответствуют последовательности MTP-CoxIV, аминокислоты 32-61 соответствуют последовательности TwinStrep, включая GG на ее С-конце, а аминокислоты 62-354 соответствуют последовательности NifH из Marichromatium gracile биотип thermosufidiphilum (Учетный №. WP_062275270).
SEQ ID NO:172. Аминокислотная последовательность полипептида MTP-CoxIV::TwinStrep::NifH, кодированная SN182. Аминокислоты 1-31 соответствуют последовательности MTP-CoxIV, аминокислоты 32-61 соответствуют последовательности TwinStrep, включая GG на ее С-конце, а аминокислоты 62-345 соответствуют последовательности NifH из Methanocaldococcus infernus (Учетный №. WP_013099459).
SEQ ID NO:173. Аминокислотная последовательность полипептида MTP-CoxIV::TwinStrep::NifH, кодированная SN183. Аминокислоты 1-31 соответствуют последовательности MTP-CoxIV, аминокислоты 32-61 соответствуют последовательности TwinStrep, включая GG на ее С-конце, а аминокислоты 62-345 соответствуют последовательности NifH из Heliobacterium modesticaldum (Учетный №. WP_012282218).
SEQ ID NO:174. Аминокислотная последовательность полипептида MTP-CoxIV::TwinStrep::NifH, кодированная SN184. Аминокислоты 1-31 соответствуют последовательности MTP-CoxIV, аминокислоты 32-61 соответствуют последовательности TwinStrep, включая GG на ее С-конце, а аминокислоты 62-335 соответствуют последовательности NifH из Chlorobium tepidum (Учетный №. WP_010933198).
SEQ ID NO:175. Аминокислотная последовательность полипептида MTP-CoxIV::TwinStrep::NifH, кодированная SN185. Аминокислоты 1-31 соответствуют последовательности MTP-CoxIV, аминокислоты 32-61 соответствуют последовательности TwinStrep, включая GG на ее С-конце, а аминокислоты 62-350 соответствуют последовательности NifH из Geobacter sp. M21 (Учетный №. WP_015837436).
SEQ ID NO:176. Аминокислотная последовательность полипептида MTP-CoxIV::TwinStrep::NifH, кодированная SN186. Аминокислоты 1-31 соответствуют последовательности MTP-CoxIV, аминокислоты 32-61 соответствуют последовательности TwinStrep, включая GG на ее С-конце, а аминокислоты 62-355 соответствуют последовательности NifH из Bradyrhizobium diazoefficans (Учетный №. AHY57040).
SEQ ID NO:177. Аминокислотная последовательность полипептида MTP-CoxIV::TwinStrep::NifH, кодированная SN187. Аминокислоты 1-31 соответствуют последовательности MTP-CoxIV, аминокислоты 32-61 соответствуют последовательности TwinStrep, включая GG на ее С-конце, а аминокислоты 62-336 соответствуют последовательности NifH из Methanobacterium thermoautotrophicum (Учетный №. AAB86034).
SEQ ID NO:178. Аминокислотная последовательность полипептида MTP-CoxIV::TwinStrep::NifH, кодированная SN188. Аминокислоты 1-31 соответствуют последовательности MTP-CoxIV, аминокислоты 32-61 соответствуют последовательности TwinStrep, включая GG на ее С-конце, а аминокислоты 62-334 соответствуют последовательности NifH из Methanosarcina (Учетный №. WP_048121466).
SEQ ID NO:179. Аминокислотная последовательность полипептида MTP-CoxIV::TwinStrep::NifH, кодированная SN189. Аминокислоты 1-31 соответствуют последовательности MTP-CoxIV, аминокислоты 32-61 соответствуют последовательности TwinStrep, включая GG на ее С-конце, а аминокислоты 62-336 соответствуют последовательности NifH из Desulfotomaculum acetoxidans (Учетный №. WP_015756624).
SEQ ID NO:180. Аминокислотная последовательность полипептида MTP-CoxIV::TwinStrep::NifH, кодированная SN190. Аминокислоты 1-31 соответствуют последовательности MTP-CoxIV, аминокислоты 32-61 соответствуют последовательности TwinStrep, включая GG на ее С-конце, а аминокислоты 62-336 соответствуют последовательности NifH из Carboxydothermus pertinax (Учетный №. WP_075859892).
SEQ ID NO:181. Аминокислотная последовательность полипептида MTP-CoxIV::TwinStrep::NifH, кодированная SN191. Аминокислоты 1-31 соответствуют последовательности MTP-CoxIV, аминокислоты 32-61 соответствуют последовательности TwinStrep, включая GG на ее С-конце, а аминокислоты 62-335 соответствуют последовательности NifH из Nostoc calcicole (Учетный №. WP_073644321).
SEQ ID NO:182. Аминокислотная последовательность полипептида MTP-FAγ51::AnfD::HA, кодированная SN81. Аминокислоты 1-54 соответствуют последовательности MTP-FAγ51, включая линкер GG на его С-конце, аминокислоты 55-572 соответствуют последовательности AnfD из A. vinelandii, а аминокислоты 573-583 соответствуют эпитопу НА.
SEQ ID NO:183. Аминокислотная последовательность полипептида HA::AnfD, кодированная SN82. Аминокислоты 1-12 соответствуют эпитопной последовательности НА, включая линкер GG на его С-конце, а аминокислоты 13-530 соответствуют последовательности AnfD из A. vinelandii.
SEQ ID NO:184. Аминокислотная последовательность полипептида MTP-FAγ51::HA::AnfK, кодированная SN129. Аминокислоты 1-53 соответствуют последовательности MTP-FAγ51, включая линкер GG на его С-конце, аминокислоты 54-64 соответствуют эпитопу HA, а аминокислоты 65-526 соответствуют последовательности AnfK из A. vinelandii.
SEQ ID NO:185. Аминокислотная последовательность полипептида MTP-FAγ51::HA::AnfH, кодированная SN130. Аминокислоты 1-53 соответствуют последовательности MTP-FAγ51, включая GG на его C-конце, аминокислоты 54-64 соответствуют эпитопу HA с GG линкером на его C-конце, а аминокислоты 65-339 соответствуют последовательности AnfH из A. vinelandii.
SEQ ID NO:186. Аминокислотная последовательность полипептида MTP-FAγ51::HA::AnfG, кодированная SN131. Аминокислоты 1-53 соответствуют последовательности MTP-FAγ51, включая GG линкер на его C-конце, аминокислоты 54-64 соответствуют эпитопу HA с GG линкером на его C-конце, а аминокислоты 65-196 соответствуют последовательности AnfG из A. vinelandii.
SEQ ID NO:187. Аминокислотная последовательность полипептида HA::AnfK, кодированная SN152. Аминокислоты 1-12 соответствуют эпитопной последовательности НА, включая линкер GG на его С-конце, а аминокислоты 13-474 соответствуют последовательности AnfK из A. vinelandii.
SEQ ID NO:188. Аминокислотная последовательность полипептида HA::AnfH, кодированная SN153. Аминокислоты 1-12 соответствуют эпитопной последовательности НА, включая линкер GG на его С-конце, а аминокислоты 13-287 соответствуют последовательности AnfH из A. vinelandii.
SEQ ID NO:189. Аминокислотная последовательность полипептида HA::AnfG, кодированная SN154. Аминокислоты 1-12 соответствуют эпитопной последовательности НА, включая линкер GG на его С-конце, а аминокислоты 13-144 соответствуют последовательности AnfG из A. vinelandii.
SEQ ID NO:190. Аминокислотная последовательность полипептида mFAγ51::HA::AnfK, кодированная SN155. Аминокислоты 1-53 соответствуют мутантной последовательности mFAγ51, включая GG линкер на ее C-конце, аминокислоты 54-64 соответствуют эпитопу HA с GG линкером на его C-конце, а аминокислоты 65-526 соответствуют последовательности AnfK из A. vinelandii.
SEQ ID NO:191. Аминокислотная последовательность полипептида mFAγ51::HA::AnfH , кодированная SN156. Аминокислоты 1-53 соответствуют мутантной последовательности mFAγ51, включая GG на ее C-конце, аминокислоты 54-64 соответствуют эпитопу HA с GG линкером на его C-конце, а аминокислоты 65-339 соответствуют последовательности AnfH из A. vinelandii.
SEQ ID NO:192. Аминокислотная последовательность полипептида mFAγ51::HA::AnfG, кодированная SN157. Аминокислоты 1-53 соответствуют мутантной последовательности mFAγ51, включая GG линкер на ее C-конце, аминокислоты 54-64 соответствуют эпитопу HA с GG линкером на его C-конце, а аминокислоты 65-196 соответствуют последовательности AnfG из A. vinelandii.
SEQ ID NO:193. Аминокислотная последовательность полипептида mFAγ51::HA::AnfD, кодированная SN158. Аминокислоты 1-53 соответствуют мутантной последовательности mFAγ51, включая GG на ее C-конце, аминокислоты 54-64 соответствуют эпитопу HA с GG линкером на его C-конце, а аминокислоты 65-582 соответствуют последовательности AnfD из A. vinelandii.
SEQ ID NO:194. Аминокислотная последовательность полипептида MTP-FAγ51::HA::AnfD, кодированная SN161. Аминокислоты 1-53 соответствуют последовательности MTP-FAγ51, включая GG линкер на его C-конце, аминокислоты 54-64 соответствуют эпитопу HA с GG линкером на его C-конце, а аминокислоты 65-582 соответствуют последовательности AnfD из A. vinelandii.
SEQ ID NO:195. Аминокислотная последовательность полипептида MTP-FAγ51::AnfD::Twin Strep, кодированная SN177. Аминокислоты 1-54 соответствуют последовательности MTP-FAγ51, включая линкер GG на его С-конце, аминокислоты 55-572 соответствуют последовательности AnfD из A. vinelandii, а аминокислоты 573-604 соответствуют эпитопу TwinStrep.
SEQ ID NO:196. Аминокислотная последовательность полипептида MTP-CoxIV::Twin Strep::AnfK, кодированная SN195. Аминокислоты 1-41 соответствуют последовательности MTP- CoxIV, включая GG линкер на его C-конце, аминокислоты 42-61 соответствуют эпитопу TwinStrep, включая GG его C-конце, а аминокислоты 62-523 соответствуют последовательности AnfK из A. vinelandii.
SEQ ID NO:197. Пептидная последовательность.
SEQ ID NO:198. Линкерная последовательность.
SEQ ID NO:199. Аминокислотная последовательность полипептида AnfD::linker16::AnfK, использованная для моделирования структуры (Пример 20). Аминокислоты 1-509 соответствуют последовательности AnfD (A. vinelandii), исключая N-концевой метионин, аминокислоты 510-525 соответствуют линкеру из 16 аминокислот, а аминокислоты 526-984 соответствуют AnfK (A. vinelandii).
SEQ ID NO:200. Линкерная последовательность.
SEQ ID NO:201. Аминокислотная последовательность полипептида AnfD::linker26(HA)::AnfK. Аминокислоты 1-517 соответствуют последовательности AnfD, аминокислоты 518-543 соответствуют линкеру из 26 аминокислот, а аминокислоты 544-1004 сответствуют AnfK.
SEQ ID NO:202. Аминокислотная последовательность полипептида MTP-FAγ51::AnfD::линкер26(HA)::AnfK, кодированная SN272. Аминокислоты 1-64 соответствуют последовательности MTP-FAγ51-HA, включая GG на ее C-конце, аминокислоты 65-581 соответствуют последовательности AnfD (A. vinelandii), аминокислоты 582-607 соответствуют линкеру из 26 аминокислот (линкер26(HA)), а аминокислоты 608-1068 соответствуют AnfK (A. vinelandii).
SEQ ID NO:203. Аминокислотная последовательность полипептида MTP-CoxIV::AnfD::линкер26(HA)::AnfK, кодированная SN273. Аминокислоты 1-61 соответствуют последовательности MTP-CoxIV, включая GG на ее C-конце, аминокислоты 62-578 соответствуют последовательности AnfD (A. vinelandii), аминокислоты 579-604 соответствуют линкеру из 26 аминокислот (линкер26(HA)), а аминокислоты 605-1065 соответствуют AnfK (A. vinelandii).
SEQ ID NO:204. Аминокислотная последовательность полипептида mFAγ51::AnfD::линкер26(HA)::AnfK, кодированная SN274. Аминокислоты 1-64 соответствуют последовательности mFAγ51, включая замещения аланина, которые не допускают расщепление MPP, и включая GG на его C-конце, аминокислоты 65-581 соответствуют последовательности AnfD (A. vinelandii), аминокислоты 582-607 соответствуют линкеру из 26-аминокислот (линкер26(HA)), а аминокислоты 608-1068 соответствуют AnfK (A. vinelandii).
SEQ ID NO:205. Аминокислотная последовательность полипептида HISx6 :: AnfD :: линкер26 (HA) :: AnfK, кодированная SN275, которая не имеет последовательности MTP и может быть локализована в цитоплазме. Аминокислоты 1-9 соответствуют последовательности HISx6, включая GG на ее C-конце, аминокислоты 10-526 соответствуют последовательности AnfD (A. vinelandii), аминокислоты 527-552 соответствуют линкеру из 26 аминокислот (линкер26(HA)), а аминокислоты 553-1013 соответствуют AnfK (A. vinelandii).
SEQ ID NO:206. Аминокислотная последовательность полипептида TbHCS (Учетный №. CP002466).
SEQ ID NO:207. Аминокислотная последовательность полипептида TpHCS (Учетный №. CP002028).
SEQ ID NO:208. Аминокислотная последовательность полипептида ScHCS (Учетный №. CP036483).
SEQ ID NO:209. Аминокислотная последовательность полипептида NsHCS (Учетный №. CP007203).
SEQ ID NO:210. Аминокислотная последовательность полипептида MaHCS (Учетный №. AE010299).
SEQ ID NO:211. Аминокислотная последовательность полипептида CtHCS (Учетный №. AE006470).
SEQ ID NO:212. Аминокислотная последовательность полипептида MiHCS1 (Учетный №. ADG13125).
SEQ ID NO:213. Аминокислотная последовательность полипептида MiHCS2 (Учетный №. ADG13175).
SEQ ID NO:214. Аминокислотная последовательность полипептида MiHCS3 (Учетный №. ADG14004).
SEQ ID NO:215. Aминокислотная последовательность полипептида LjFEN1 (Учетный №. BAI49592).
SEQ ID NO:216. Аминокислотная последовательность AnfD из A. vinelandii (Учетный №. WP_012703361); 518aa.
SEQ ID NO:217. Аминокислотная последовательность AnfK из A. vinelandii (Учетный №. WP_012703359); 462aa.
SEQ ID NO:218. Аминокислотная последовательность AnfH из A. vinelandii (Учетный №. WP_012703362); 275aa.
SEQ ID NO:219. Аминокислотная последовательность AnfG из A. vinelandii (Учетный №. WP_012703360); 132aa.
SEQ ID NO:220. Пептидная последовательность.
SEQ ID NO:221. Аминокислотная последовательность из N. benthamiana P72026; 606aa.
SEQ ID NO:222. Аминокислотная последовательность из N. benthamiana P20586; 470aa.
SEQ ID NO:223. Аминокислотная последовательность из Mycobacterium tuberculosis α-изопропилмалатсинтазы (MtLeuA); 644aa.
SEQ ID NO:224; Аминокислотная последовательность полипептида NifH из A. vinelandii (AvNifH; Учетный №.WP_012698831); 290aa.
SEQ ID NO:225. Пептидная последовательность AnfH motif I, где X представляет собой любую аминокислоту.
SEQ ID NO:226. Пептидная последовательность AnfH motif II.
SEQ ID NO:227. Пептидная последовательность AnfH motif III.
SEQ ID NO:228. Пептидная последовательность AnfH motif IV.
SEQ ID NO:229. Пептидная последовательность AnfH motif V, где X представляет собой любую аминокислоту.
SEQ ID NO:230. Пептидная последовательность AnfH motif VI.
SEQ ID NO:231. Пептидная последовательность AnfH motif VII, где X представляет собой любую аминокислоту.
SEQ ID NO:232. Аминокислотная последовательность FdxN белка из A. vinelandii; Учетный №. WP_012703542; 92aa.
SEQ ID NO:233. Аминокислотная последовательность слитого полипептида MTP-FAγ51-FdxN-HA из SN291; 157aa. Аминокислоты 1-54 соответствуют последовательности MTP-FAγ51 с линкером GG, аминокислоты 55-145 соответствуют последовательности FdxN без N-концевого метионина, а аминокислоты 146-157 соответствуют эпитопу НА.
SEQ ID NO:234. Аминокислотная последовательность слитого полипептида MTP-FAγ51-HA-FdxN из SN292; 156aa. Аминокислоты 1-53 сответствуют последовательности MTP-FAγ51 с линкером GG, аминокислоты 54-64 соответствуют эпитопу HA с линкером GG, а аминокислоты 65-156 соответствуют последовательности FdxN без N-концевого метионина.
SEQ ID NO:235. Аминокислотная последовательность слитого полипептида mFAγ51-HA-FdxN из SN299; 156aa. Аминокислоты 1-53 сответствуют последовательности mFAγ51 с линкером GG, аминокислоты 54-64 соответствуют эпитопу HA с линкером GG, а аминокислоты 65-156 соответствуют последовательности FdxN без N-концевого метионина.
SEQ ID NO:236. Аминокислотная последовательность слитого полипептида HA-FdxN из SN300; 104aa. Аминокислоты 1-12 соответствуют эпитопу HA с линкером GG, а аминокислоты 13-104 соответствуют последовательности FdxN без N-концевого метионина.
SEQ ID NO:237. Аминокислотная последовательность слитого полипептида MTP-FAγ51-HA-NifV из SN254; 448aa. Аминокислоты 1-53 соответствуют последовательности MTP-FAγ51 с линкером GG, аминокислоты 54-64 соответствуют эпитопу HA с линкером GG, а аминокислоты 65-448 соответствуют последовательности NifV из A. vinelandii.
SEQ ID NO:238. Аминокислотная последовательность полипептида NafY из A. vinelandii (AvNafY; Учетный №. AGK13761).
SEQ ID NO:239. C-концевая аминокислотная последовательность полипептида NifK.
SEQ ID NO:240. C-концевая аминокислотная последовательность полипептида NifK.
SEQ ID NO:241. C-концевая аминокислотная последовательность полипептида NifK.
SEQ ID NO:242. C-концевая аминокислотная последовательность полипептида NifK.
SEQ ID NO:243. C-концевая аминокислотная последовательность полипептида NifK.
SEQ ID NO:244. C-концевая аминокислотная последовательность полипептида AnfK.
SEQ ID NO:245. C-концевая аминокислотная последовательность полипептида AnfK.
SEQ ID NO:246. C-концевая аминокислотная последовательность полипептида AnfK.
SEQ ID NO:247. C-концевая аминокислотная последовательность полипептида AnfK.
SEQ ID NO:248. C-концевая аминокислотная последовательность полипептида AnfK.
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Общие способы и определения
Все технические и научные термины, используемые в данном документе, если специально не указано иное, должны иметь такое же значение, как оно обычно понимается специалистом в данной области науки (напр., в культуре клеток, молекулярной генетике, молекулярной биологии растений, химии белков и биохимии).
Рекомбинантный белок, культура клеток и иммунологические методы, используемые в настоящем изобретении, если не указано иное, являются стандартными процедурами, хорошо известными специалистам в данной области. Эти способы описаны и разобраны в литературных источниках: J. Perbal, A Practical Guide to Molecular Cloning, John Wiley и Sons (1984), J. Sambrook et al., Molecular Cloning: A Laboratory Manual, Cold Spring Harbour Laboratory Press (1989), T.A. Brown (editor), Essential Molecular Biology: A Practical Approach, Volumes 1 и 2, IRL Press (1991), D.M. Glover и B.D. Hames (editors), DNA Cloning: A Practical Approach, Volumes 1-4, IRL Press (1995 и 1996), и F.M. Ausubel et al. (editors), Current Protocols in Molecular Biology, Greene Pub. Associates и Wiley-Interscience (1988, including all updates until present), Ed Harlow и David Lane (editors) Antibodies: A Laboratory Manual, Cold Spring Harbour Laboratory, (1988), и J.E. Coligan et al. (editors) Current Protocols in Immunology, John Wiley & Sons (включая все последние обновления).
Термин «и/или», например, «X и/или Y» следует понимать как означающий «X и Y» или «X или Y», и явную поддержку обоих значений или любого значения.
В контексте данного документа, термин «примерно», если не указано иное, относится к значению, составляющему +/- 10%, или более предпочтительно +/- 5% от заданного значения.
В данном описании слово «содержать» или его варианты, такие как «содержит» или «содержащий», подразумевает включение указанного элемента, целого числа или шага или группы элементов, целых чисел или шагов, но не исключает любого другого элемента, целого числа или шага, или группы элементов, целых чисел или шагов.
Нитрогеназа
Нитрогеназа представляет собой фермент эубактерии и архебактерии, который катализирует восстановление прочной тройной связи азота (N2) с образованием аммиака (NH3). В природе нитрогеназа содержится только в бактериях. Это комплекс двух ферментов, которые можно очищать по отдельности, а именно динитрогеназой и редуктазой динитрогеназы. Динитрогеназа, также называемая компонентом I или молибден-железным (MoFe) белком, представляет собой тетрамер из двух полипептидов NifD и двух полипептидов NifK (α2β2), который также содержит два «P-кластера» и два «FeMo-кофактора» (FeMo-ко). Каждая пара субъединиц NifD-NifK содержит один P-кластер и один FeMo-ко. FeMo-ко представляет собой металлокластер, состоящий из кластера MoFe3-S3 в комплексе с гомоцитратной молекулой, которая координирована с атомом молибдена и соединена мостиком с кластером Fe4-S3 тремя лигандами серы. FeMo-ко собирается отдельно в клетках и затем встраивается в белок апо-MoFe. P-кластер также является металлокластером и содержит 8 атомов Fe и 7 атомов серы со структурой, аналогичной, но отличной от FeMo-кo. P-кластеры локализуются на границе αβ-субъединицы динитрогеназы и координируются цистеинильными остатками из обеих субъединиц. Редуктаза динитрогеназы, также называемая компонентом II или «белком Fe», представляет собой димер полипептидов NifH, который также содержит один кластер Fe4-S4 на границе раздела субъединицы и два сайта связывания Mg-ATP, по одному на каждую субъединицу. Этот фермент является обязательным донором электронов для динитрогеназы, где электроны передаются от кластера Fe4-S4 к P-кластеру и, в свою очередь, к FeMo-ко, сайту для восстановления N2.
Хотя Mo-содержащая нитрогеназа является наиболее часто встречающейся нитрогеназой в бактериях, также существуют две гомологичные нитрогеназы, которые генетически различаются, но имеют сходные составы кофакторов и субъединиц, а именно ванадийсодержащую нитрогеназу и только Fe-нитрогеназу, кодированную генами Vnf (ванадиевая азотфиксация ) и Anf (альтернативная азотфиксация), соответственно. В природе некоторые бактерии обладают всеми тремя типами нитрогеназ, другие бактерии содержат только Mo- и V-содержащие ферменты или только Mo-содержащий фермент, например Klebsiella pneumoniae.
Для биосинтеза FeMo-ко и созревания компонентов нитрогеназы до их каталитически активных форм требуются различные гены азотфиксации (Nif). В работе (Rubio и Ludden, 2008) описаны роли полипептидов NifB, NifE, NifH, NifN, NifQ, NifV и NifX в синтезе FeMo-ко.
Биологическая фиксация N2, катализируемая прокариотическим ферментом нитрогеназы, является альтернативой использованию синтетических N2 удобрений. Чувствительность нитрогеназы к кислороду является основным препятствием для инженерии биологической фиксации азота в растениях, например, в зерновых культурах, путем прямого переноса гена Nif.
Авторы настоящего изобретения считают, что нацеливание полипептидов Nif на митохондриальный матрикс (ММ) растительных клеток может преодолеть проблему чувствительности к кислороду. ММ содержит ферменты, потребляющие кислород, которые позволяют функционировать другим ферментам, содержащим чувствительный к кислороду кластер Fe-S. Механизм сборки митохондриальных кластеров Fe-S подобен сборке диазотрофных эквивалентов (Balk and Pilon, 2011; Lill and Mühlenhoff, 2008). Следовательно, некоторые из требований для биосинтеза нитрогеназы могут уже присутствовать в ММ, уменьшая количество генов Nif, необходимых для реконструкции. Также существует высокий восстановительный потенциал и высокая концентрация ATP (Geigenberger and Fernie, 2014; Mackenzie and McIntosh, 1999), что является предпосылкой для ферментативного катализа нитрогеназы. Кроме того, присутствие глутаматсинтазы в митохондриях обеспечивает точку входа для любого аммиака, фиксированного нитрогеназой, для участия в метаболизме растения. Учитывая эти характеристики и тот факт, что сами митохондрии имеют α-протеобактериальное происхождение, авторы настоящего изобретения сочли, что данная органелла хорошо подходит в качестве места для попытки функциональной реконструкции нитрогеназы.
В качестве первого шага к реконструкции нитрогеназы в митохондриях растительной клетки были необходимы доказательства того, что отдельные белки Nif могут быть правильно нацелены на ММ. С этой целью изобретатели выбрали модельное растение Nicotiana benthamiana в качестве платформы экспрессии (Wood et al., 2009), чтобы обеспечить экспрессию трансгенов по отдельности или, что более важно, в комбинациях. Поскольку большинство белков, локализованных в ММ, кодируются ядром, авторы настоящего изобретения полагались на недавние достижения в понимании субклеточной связи и процесса транспорта (Huang et al., 2009; Murcha et al., 2014), с использованием ранее охарактеризованного нацеливающего сигнала N-концевого пептида (Lee et al., 2012).
Модельный бактериальный диазотроф Klebsiella pneumoniae использует 16 уникальных белков для биосинтеза и каталитической функции нитрогеназы. Авторы настоящего изобретения реконструировали все 16 белков Nif из K. pneumoniae для нацеливания на ММ растения и оценили их экспрессию и процессинг в листьях N. benthamiana. Все 16 полипептидов Nif были транзиентно экспрессированы и исследованы для процессинга специфичной последовательности ММ. Авторы настоящего изобретения установили, что все 16 полипептидов Nif могут экспрессироваться по отдельности, как слитые полипептиды MTP:Nif в клетках листьев растений. Кроме того, авторы настоящего изобретения предоставляют доказательства того, что эти белки могут быть нацелены на митохондриальный матрикс (MM), субклеточную локализацию, потенциально способствующую выполнению функции нитрогеназы, и могут быть расщеплены митохондриальной процессинговой протеазой (MPP). Это является важным прогрессом для достижения цели создания эндогенной фиксации азота в растениях.
Импорт митохондриального белка в растениях
Почти все митохондриальные белки кодируются ядром и транслируются в цитозоле, поэтому требуется их транслокализация в митохондриях. Сигнальные последовательности внутри полипептидов направляют их импорт в четыре различных внутри-митохондриальных локализации: внешнюю мембрану (OM), внутримембранное пространство (IS), внутреннюю мембрану (IM) или матрикс (MM). Эти сигнальные последовательности различаются по своим биохимическим свойствам и направляют миграцию, по меньшей мере, через четыре различных пути импорта, которые направляют полипептиды в одну или несколько из четырех локализаций (Chacinska et al., 2009). Четыре пути представляют собой: (1) общий путь импорта, также называемый «классическим» путем препоследовательности, который направляет полипептиды к MM, IS или IM; (2) путь импорта носителем, используемый для транспорта в IM; (3) путь сборки митохондриального внутримембранного пространства (MIA); и (4) путь механизма сортировки и сборки аппарата (SAM), используемый для транспорта полипептидов в OM. Общим путем импорта импортируются полипептиды, имеющие расщепляемую препоследовательность, также известную как сигнальная последовательность. Эти полипептиды также могут иметь сигнал гидрофобной сортировки (HSS). Путем импорта носителем импортируются полипептиды с внутренними сигналами, подобными препоследовательности, и с гидрофобной областью. Путем MIA импортируются полипептиды с двойными цистеиновыми остатками. Путем SAM импортируются полипептиды, которые содержат сигнал β и предполагаемый сигнал TOM20. Все эти пути используют транслоказу внешней мембраны (TOM), а первый и второй пути также используют транслоказу TIM23 внутримембранного комплекса. Только в первом пути используется пептидаза матричного процессинга (протеаза матричного процессинга, MPP).
Общей характеристикой всех полипептидов, нацеленных на митохондрии, является присутствие, по меньшей мере, одного домена в полипептиде, который направляет транспорт в правильную локализацию. Наиболее изученным из них является «классический» N-концевой домен препоследовательности, который расщепляется в матриксе с помощью MPP (Murcha et al., 2004). Было подсчитано, что около 70% митохондриальных белков растений и животных имеют расщепляемую препоследовательность, но также были обнаружены, как внутренние, так и С-концевые сигнальные последовательности (см. Pfanner и Geissler (2001), Schleiff и Soll (2000)). В Arabidopsis, этот диапазон препоследовательностей имеют длину от 11 до 109 аминокислотных остатков при средней длине в 50 аминокислотных остатков. Хотя не существует обобщающей типичной последовательности, которая полностью формирует препоследовательность для первого пути, они, как правило, содержат высокую долю гидрофобных и положительно заряженных аминокислот. Дополнительной характеристикой является их способность образовывать амфифильную α-спираль, обычно начиная с первых 10 аминокислотных остатков (Roise et al., 1986). Эти домены богаты гидрофобными (Ala, Leu, Phe, Val), гидроксилированными (Ser, Thr) и положительно заряженными (Arg, Lys) аминокислотными остатками, а также не содержат кислых аминокислот. В большом количестве митохондриальных белков серин (16-17%) и аланин (12-13%) чрезмерно представлены в митохондриальных сигнальных пептидах, а аргинин присутствует в избытке (12%). Точка расщепления МРР формируется для большинства препоследовательностей в результате присутствия консервативного остатка аргинина, обычно в позиции Р2 (-2 аa от расщепляющей связи), или Р3 в большинстве других случаев. (Huang et al., 2009).
Митохондриальные препоследовательности взаимодействуют с рецептором Tom20 через гидрофобные остатки. Исследования показали, что гидрофобная поверхность α-спирали способствует распознаванию пептида компонентом TOM20 импортного комплекса TOM, тогда как положительные заряды распознаются субъединицей TOM22 (Abe et al., 2000). Наконец, большинство препоследовательностей направляют транспорт полипептида в ассоциации с Hsp70, и, соответственно, почти все препоследовательности растений содержат, по меньшей мере, один связывающий мотив для молекулярного шаперона Hsp70 (Zhang и Glaser, 2002). Шаперон Hsp70 участвует в укладывании белка, предотвращает агрегацию белков и действует как молекулярный двигатель, протягивая предшественник через митохондриальные мембраны. Электрический трансмембранный потенциал (Δψ) (~ 100 мВ, отрицательный внутри) также управляет через внутреннюю мембрану транслокализацией положительно заряженной препоследовательности, посредством электрофоретического эффекта.
Большинство белков с расщепляемыми препоследовательностями предназначены для митохондриального матрикса через общий путь импорта, который использует переносчик комплекса внешней мембраны (TOM) и переносчик комплекса внутренней мембраны 23 (TIM23). Однако некоторые белки с расщепляемыми препоследовательностями могут собираться во внутренней мембране (Murcha et al., 2004) или в внутримембранном пространстве, если они также содержат сигнал гидрофобной сортировки (HSS)(Glick et al., 1992). Существует очень мало примеров белков, локализованных в матриксе, у которых нет их расщепленных препоследовательностей. В матриксе Arabidopsis была обнаружена только глутаматдегидрогеназа с непроцессированной полноразмерной препоследовательностью. (Huang et al., 2009).
Для белков, которые не нацелены на матрикс, используются различные сигналы для внутренней нерасщепляемой локализации. Они обычно связаны с специфичным путем миграции и дополнительно адаптированы для конкретного класса белков. В растениях до сих пор не определено, что именно составляет внутреннюю сигнальную последовательность для белков внутримембранного пространства. Однако, похоже, что мотив с двойными цистеиновыми остатками связан с переносом через митохондриальный путь сборки внутримембранного пространства (MIA) (Carrie et al., 2010; Darshi et al., 2012). Наконец, нерасщепляемые внутренние последовательности также используются белками, предназначенными для внутренней мембраны через путь носителя, который использует механизм TOM и TIM22 для вставки белков с множеством трансмембранных областей (Kerscher et al., 1997; Sirrenberg et al., 1996). Эти последовательности обычно содержат гидрофобную область, за которой следует препоследовательность, подобная внутренней последовательности, и, таким образом, подобная N-концевым препоследовательностям, но отличающаяся своей внутренней локализацией в родственном им белке.
В фотосинтезирующих организмах, кодируемые ядром митохондриальные белки, необходимы для дифференциации хлоропластов и митохондриальной миграции, несмотря на многие сходства между этими двумя органеллами и их протеомами. Данная α-спираль, которая встречается в основном в препоследовательностях митохондрий, обычно отсутствует в препоследовательностях хлоропластов (Zhang and Glaser, 2002), которые имеют тенденцию быть более неструктурированными и демонстрируют доменную структуру с высоким содержанием β-складок (Bruce, 2001).
В растениях MPP закрепляется на связанном с внутренней мембраной комплексе Cytbc1, хотя активный сайт MPP локализуется напротив матрикса, а функции двух белков остаются независимыми. (Glaser and Dessi, 1999).
Митохондриальный нацеливающий пептид
В контексте данного документа, термин «митохондриальный нацеливающий пептид» или «MTP» означает аминокислотную последовательность, содержащую, по меньшей мере, 10 аминокислот и предпочтительно от 10 до примерно 80 аминокислотных остатков в длину, которая направляет целевой белок в митохондрии, и которая может использоваться гетерологично при трансляционном слиянии целевого белка МТР для направления выбранного целевого белка, например, полипептида Nif, Gus, GFP и т. д., в митохондрии.
MTP обычно содержит на своем N-конце инициатор трансляции метионина полипептида, из которого он получен. MTP трансляционно слит с полипептидом Nif или «целевым белком» посредством пептидной связи с остатком Met, который соответствует инициатору Met целевого белка, или этот остаток Met может быть опущен, а пептидная связь непосредственно слита с аминокислотным остатком, который является второй аминокислотой целевого белка у дикого типа. MTP обычно богат основными и гидроксилированными аминокислотами и обычно не имеет кислых аминокислот или протяженных гидрофобных участков. MTP может образовывать амфифильные спирали.
Не вдаваясь в теорию, MTP обычно содержит последовательность, нацеленную на поглощение, которая связывается с рецепторами на внешней мембране митохондрий. После связывания с внешней мембраной слитой полипептид предпочтительно подвергается мембранной транслокализации, для переноса канальных белков и прохождения через двойную мембрану митохондрий в митохондриальный матрикс (ММ). Затем последовательность, нацеленная на поглощение, обычно расщепляется, а зрелый слитой белок укладывается.
MTP может содержать дополнительные сигналы, которые впоследствии нацеливают белок в различные области митохондрий, например, в митохондриальный матрикс (MM). В одном из вариантов осуществления, последовательность, нацеленная на поглощение, представляет собой последовательность нацеливания на матрикс.
MTP может быть расщепляема или нерасщепляема при трансляционном слиянии с полипептидом Nif. Таким образом, в одном из вариантов осуществления, слитой полипептид MTP-Nif, по меньшей мере, частично расщеплен. В этом отношении фраза «по меньшей мере частично расщеплен» относится к детектируемой величине расщепления слитого полипептида MTP-Nif при экспрессии в растительной клетке. В одном из вариантов осуществления, по меньшей мере 50% слитого полипептида MTP-Nif, который продуцируется в клетке, расщепляется внутри последовательности MTP, предпочтительно расщепляется, по меньшей мере, 75%, более предпочтительно расщепляется, по меньшей мере, 90%. В альтернативном варианте осуществления, менее 50% слитого полипептида MTP-Nif расщепляется в клетке, например, MTP не расщепляется. В одном из вариантов осуществления, MTP не содержит сайта расщепления для MPP. MTP может содержать сайт расщепления. При расщеплении N-концевая часть полученного процессированного продукта (т. е. зрелый NP) может содержать одну или несколько C-концевых аминокислот MTP, также называемых здесь последовательностью scar, или не scar последовательностью. При наличии scar последовательность предпочтительно имеет длину от 1 до 45 аминокислот, более предпочтительно от 1 до 20 аминокислот, еще более предпочтительно от 1 до 12 аминокислот. Альтернативно сайт расщепления может локализоваться внутри слитого полипептида таким образом, что вся последовательность MTP отщепляется, например, линкер может содержать последовательность расщепления.
Нативные митохондриальные нацеливающие пептиды локализуются на N-конце белков-предшественников, а N-концевая часть обычно отщепляется во время или после импорта в митохондрии. Расщепление обычно катализируется протеазой матричного процессинга (MPP), которая интегрирована в комплекс bc1 дыхательной цепи растений. Эта протеаза распознает сайты расщепления почти 1000 белков-предшественников, которые имеют широкий спектр аминокислотных последовательностей и которые мало консервативны. В одном из вариантов осуществления, MTP содержит сайт расщепления протеазой для MPP. В другом варианте осуществления, процессированный продукт получают путем расщепления посредством MPP слитого белка внутри или сразу после MTP. В контексте данного документа, фраза «сразу после» означает, что после расщепления с помощью MPP не остается аминокислот от MTP, слитого с полипептидом Nif. Таким образом, когда слитой полипептид расщепляется «сразу после» MTP, сайт расщепления MPP находится сразу после C-концевой аминокислоты MTP.
Термины « расщепленный продукт» или «продукт расщепления», используемые здесь в контексте слитого полипептида MTP, относятся к полипептиду, полученному в результате расщепления протеазой внутри или сразу после аминокислотной последовательности MTP. В этом отношении продукт расщепления слитого полипептида MTP может быть получен путем расщепления с помощью MPP. Расщепленный продукт может сохранять одну или несколько аминокислот из MTP после расщепления (т. е. пептид scar) или он может не иметь никаких аминокислот, оставшихся от MTP после расщепления. В одном из вариантов осуществления, расщепленный продукт слитого полипептида Nif, согласно изобретению, содержит, по меньшей мере, 95% или все аминокислоты, присутствующие в последовательности полипептида Nif.
В одном из вариантов осуществления, MTP не расщепляется. Авторы настоящего изобретения продемонстрировали, что встраивание MTP не всегда приводило к полному процессингу белков Nif.. В некоторых случаях (NifX-FLAG, NifD-HAopt1 и NifDK-HA) наблюдались как процессированные, так и непроцессированные белки Nif. Учитывая, что не существует обобщающей типичной последовательности для MTP, а внутренние белковые последовательности могут влиять на митохондриальное нацеливание(Becker et al., 2012), возможно, что неудивительно, авторы настоящего изобретения обнаружили различия в эффективности процессинга среди белков Nif.
Подходящие MTP, которые можно использовать в контексте настоящего изобретения, содержат, не ограничиваясь, пептиды, имеющие общую структуру, определенную в работах von Heijne (1986) или Roise и Schatz (1988). Неограничивающими примерами MTP являются митохондриальные нацеливающие пептиды, представленные в Таблице I работы von Heijne (1986) или описанные здесь.
В одном из вариантов осуществления, MTP представляет собой γ-субъединицу F1-ATPase (MTP-FAγ). Примером подходящего FAγ MTP является таковой из A. thaliana (Lee et al., 2012). В одном из вариантов осуществления, MTP-FAγ имеет в длину 77 аминокислот, которые при расщеплении MMP, оставляет 35 остатков MTP в N-концевой области слитого полипептида. В предпочтительном варианте осуществления MTP-FAγ имеет в длину менее 77 аминокислот. Например, MTP-FAγ может иметь в длину примерно 51 аминокислоту, которые при расщеплении MMP, оставляет 9 остатков MTP в N-концевой области слитого полипептида.
Квалифицированному специалисту очевидно, что существует программное обеспечение для прогнозирования митохондриальных белков и их нацеливающей последовательности, например MitoProtII, PSORT, TargetP и NNPSL.
MitoProtII представляет собой программу, которая прогнозирует митохондриальную локализацию последовательности на основе нескольких физико-химических параметров (напр., аминокислотного состава в N-концевой части или максимальной общей гидрофобности для окна из 17 остатков). PSORT представляет собой программу, которая предсказывает субклеточную локализацию на основе различных характеристик, производных от последовательностей, например, наличия мотивов последовательностей и аминокислотных составов. Программа TargetP предсказывает субклеточную локализацию эукариотических белков на основе предсказанного присутствия любой из N-концевых предпоследовательностей: транзитного пептида хлоропласта, митохондриального нацеливающего пептида или сигнального пептида секреторного пути. Для программы TargetP требуется N-концевая последовательность для входа в два уровня искусственных нейронных сетей (ANN), использующих более ранние бинарные предикторы, SignalP и ChloroP. Для последовательностей, которые, как предсказано, содержат N-концевую препоследовательность, также может быть предсказан потенциальный сайт расщепления. Программа NNPSL использует еще один способ на основе ANN, использующий аминокислотный состав для определения одной из четырех субклеточных локализаций (цитозольной, внеклеточной, ядерной и митохондриальной) искомой последовательности.
Квалифицированный специалист способен легко определить, нацелил ли, выбранный MTP, слитой полипептид на митохондриальный матрикс, на основе обычных способов и способов, описанных в данном документе. Авторы настоящего изобретения выбрали нацеливающий пептид, ранее продемонстрированный как способный переносить GFP в протопластах Arabidopsis, (Lee et al., 2012), который является относительно длинным, чтобы способствовать обнаружению процессированного белка. Как показано в приведенных здесь Примерах, выбранный MTP нацелил все выбранные белки нитрогеназы на MM. Этот вывод основан на нескольких доказательствах. Во-первых, размеры, наблюдаемые для полипептидов Nif, экспрессируемых N. benthamiana, соответствовали ожидаемому размеру, полученному в результате процессинга ММ-пептидазой. Это также отразилось на отклонении в размере, наблюдаемом между бактериальными (полноразмерные непроцессированные) и митохондриальными белками растений, экспрессированными Nif небольшого размера (NifF и NifZ). Кроме того, мутация MTP, делающая его неспособным к процессингу механизмом импорта митохондрий, приводила к образованию большей полосы для слияний NifD и GFP, что согласуется с отклонениями в размере между и непроцессированным белком. Наконец, методом масс-спектрометрии для типичного слитого полипептида было определено, что MTP-NifH был расщеплен между остатками 42-43 MTP, как предсказано для специфичного процессинга в матриксе.
В некоторых вариантах осуществления этого изобретения, полезно использовать несколько тандемных копий выбранного MTP. Кодирующую последовательность для дублированного или размноженного нацеливающего пептида можно получать с помощью генной инженерии из существующего MTP. Количество MTP может быть измерено клеточным фракционированием с последующим, например, количественным иммуноблот-анализом. Таким образом, в настоящем изобретении, термин «митохондриальный нацеливающий пептид» или «MTP» охватывает одну или несколько копий одного аминокислотного пептида, который нацеливает целевой белок Nif в митохондрии. В предпочтительном варианте осуществления, MTP содержит две копии выбранного MTP. В другом варианте осуществления MTP содержит три копии выбранного MTP. В другом варианте осуществления MTP содержит четыре или более копий выбранного MTP.
Специалисту очевидно, что последовательность MTP не ограничивается нативными последовательностями MTP, но может содержать аминокислотные замены, делеции и/или вставки по сравнению с природным MTP, при условии, что вариант последовательности по-прежнему функционирует для митохондриального нацеливания.
Специалисту очевидно, что MTP может быть фланкирована аминокислотами в его N- или C-концевых областях в результате стратегии клонирования и может функционировать как линкер. Эти дополнительные аминокислоты можно рассматривать как часть MTP.
Специалисту очевидно, что MTP может быть слит на N- или C-конце с олигопептидным линкером и/или меткой, например, меткой эпитопа. В предпочтительном варианте осуществления, один или несколько или все слитые полипептиды Nif, согласно изобретению, продуцированные в растительной клетке, не имеют добавленных меток эпитопа по сравнению с соответствующим полипептидом Nif дикого типа.
Митохондриальный нацеливающий пептид (MTP)-слитые полипетиды Nif
Настоящее изобретение относится к слитым полипептидам Nif митохондриального нацеливающего пептида (MTP) и их расщепленным полипептидным продуктам. Когда, согласно изобретению, слитой полипептид MTP-Nif экспрессируется в растительной клетке, то либо слитой полипептид MTP-Nif и/или продукт расщепленного полипептида нацеливается на митохондриальный матрикс (MM). Предпочтительно, слитые полипептиды придают нитрогеназно-редуктазную и/или нитрогеназную активность растительной клетке или активность, которая является такой же, как та, которую придает соответствующий полипептид Nif дикого типа в бактерии.
В контексте данного документа, термин «слитой полипептид» означает полипептид, который содержит два или более полипептидных домена, ковалентно связанных пептидной связью. Обычно, согласно изобретению, слитой полипептид кодируется в виде одной полипептидной цепи химерным полинуклеотидом. В одном из вариантов осуществления, слитые полипептиды согласно изобретению содержат митохондриальный нацеливающий пептид (MTP) и полипептид Nif (NP). В этом варианте осуществления, C-концевая область MTP трансляционно слита с N-концевой областью NP. В альтернативном варианте осуществления, слитые полипептиды, согласно изобретению, содержат С-концевую часть MTP и NP, где C-концевая часть является результатом расщепления MTP с помощью MPP. Такая C-концевая часть MTP упоминается здесь как последовательность «scar». В этом варианте осуществления, C-концевая аминокислота C-концевой части MTP трансляционно сливается с N-концевой аминокислотой NP. В этих вариантах осуществления, слитой полипептид может содержать одну или несколько дополнительных аминокислот между MTP и NP, например последовательность GlyGly, и/или добавленный метионин в качестве аминокислоты начала трансляции. В одном варианте осуществления, слитой полипептид содержит два полипептида Nif, предпочтительно, полипептид NifD, трансляционно слитой через линкерную последовательность с полипептидом NifK, или полипептид NifE, трансляционно слитой через линкерную последовательность с полипептидом NifN. Могут присутствовать оба этих слитых полипептида. В этих вариантах осуществления, предпочтительно, чтобы второй полипептид Nif в слитом полипептиде имел свой С-конец дикого типа, т.е. не имел какого-либо С-концевого продолжения.
В контексте данного документа, термин «трансляционно слитой в N-концевой области» означает, что C-концевая область полипептида MTP или линкерного полипептида ковалентно присоединена пептидной связью к N-концевой области NP, тем самым являясь слитым полипептидом. В одном из вариантов осуществления, NP не содержит свой нативный остаток начала трансляции метионина (Met) или два своих N-концевых остатка Met по сравнению с соответствующим NP дикого типа. В альтернативном варианте осуществления, NP содержит начало трансляции Met или один или оба из двух N-концевых остатков Met полипептида NP дикого типа, например, для NifD.
Такие полипептиды обычно продуцируется путем экспрессии кодирующей химерный белок области, где трансляционная рамка считывания нуклеотидов, кодирующих MTP, соединена в рамке с рамкой считывания нуклеотидов, кодирующих NP. Специалисту очевидно, что C-концевая аминокислота MTP может быть трансляционно слита с N-концевой аминокислотой NP без линкера или через линкер из одного или нескольких аминокислотных остатков, например, 1-5 аминокислотных остатков. Таким образом, линкер может рассматриваться как часть MTP. Экспрессия кодирующей белок области, может сопровождаться расщеплением MTP в ММ растительной клетки, а такое расщепление (если оно происходит) включено в концепцию получения слитого полипептида согласно изобретению.
Слитой полипептид или процессированный полипептид Nif предпочтительно обладает функциональной активностью Nif. В предпочтительном варианте осуществления, данная активность аналогична активности соответствующего полипептида Nif дикого типа. Функциональная активность слитого полипептида или процессированного полипептида Nif может быть определена в анализах бактериальной и биохимической комплементации. В предпочтительном варианте осуществления, слитой полипептид или процессированный полипептид Nif имеет примерно 70-100% активности Nif дикого типа. Полипептиды Nif, не обладающие функциональностью Nif, по-прежнему могут использоваться, например, в качестве исследовательских инструментов для проверки уровней экспрессии генетических конструкций или для ассоциации с другими полипептидами Nif.
Слитой полипептид может содержать более одного MTP и/или более одного NP, например, слитой полипептид может содержать MTP, полипептид NifD и полипептид NifK. Слитой полипептид может также содержать олигопептидный линкер, например, связывающий два NP. Предпочтительно линкер имеет достаточную длину, чтобы позволить двум или более функциональным доменам, например, двум NP, например, NifD и NifK или NifE и NifN, связываться по функциональной схеме в растительной клетке. В предпочтительных вариантах осуществления, полипептид NifD представляет собой полипептид AnfD, а полипептид NifK представляет собой полипептид AnfK. Такой линкер может иметь в длину от 8 до 50 аминокислотных остатков, предпочтительно, примерно 25-35 аминокислот в длину, более предпочтительно, примерно 30 аминокислотных остатков в длину или примерно 26 аминокислотных остатков в длину для слитого полипептида AnfD-линкер-AnfK. Слитой полипептид можно получать обычными способами, напр., посредством экспрессии гена полинуклеотидной последовательности, кодирующей указанный слитой полипептид, в подходящей клетке.
В контексте данного документа, термин «существу очищенный полипептид» означает полипептид, который по существу не содержит компонентов (напр., липидов, нуклеиновых кислот, углеводов), которые обычно связываются с полипептидом, например, в клетке. Предпочтительно, по существу очищенный полипептид, по меньшей мере, на 90% свободен от указанных компонентов.
Растительные клетки, трансгенные растения и их части, согласно изобретению, содержат полинуклеотид, кодирующий полипептид, согласно изобретению. Полипептиды, согласно изобретению, не встречаются в природе в растительных клетках, в частности, не в митохондриях растительных клеток, и поэтому полинуклеотид, кодирующий полипептид, может упоминаться здесь как экзогенный полинуклеотид, поскольку он не встречается в растительной клетке в природе, но вводился в растительную клетку или в клетку-предшественницу. Клетки, растения и части растений, согласно изобретению, которые продуцируют полипептид согласно изобретению, продуцируют рекомбинантный полипептид. В контексте данного документа, термин «рекомбинантный» полипептид относится к полипептиду, кодированному экзогенным полинуклеотидом при продуцировании клеткой, причем этот полинуклеотид был введен в клетку или клетку-предшественницу с помощью методов рекомбинантной DNA или RNA, например, трансформацией. Обычно растительная клетка, растение или часть растения содержит неэндогенный ген, который вызывает продуцирование определенного количества полипептида, по крайней мере, в какой-то момент жизненного цикла растительной клетки или растения. Предпочтительно, экзогенный полинуклеотид интегрируется в ядерный геном растительной клетки и/или транскрибируется в ядре клетки.
В одном из вариантов осуществления, полипептид, согласно изобретению, не является полипептидом природного происхождения. В альтернативном варианте осуществления, полипептид, согласно изобретению, встречается в природе, но присутствует в растительной клетке, предпочтительно в митохондриях растительной клетки, в которой он не встречается в природе.
В одном из вариантов осуществления, полипептид, согласно изобретению, (напр., слитой полипептид MTP или его расщепленный продукт), по меньшей мере, частично растворим в митохондриях растительной клетки. В контексте данного документа, фраза «по меньшей мере, частично растворим» означает, что полипептид обнаруживается в растворимой фракции гомогенизированного образца, содержащей митохондрии растительной клетки. Подходящие способы определения растворимости полипептидов известны в области техники, к которой относится данное изобретение, и включают те, которые описаны в Примере 1. В одном из вариантов осуществления, по меньшей мере, 5%, по меньшей мере, 10%, по меньшей мере, 15%, по меньшей мере, 20%, по меньшей мере, 25%, по меньшей мере, 30%, по меньшей мере, 40%, по меньшей мере, 50%, по меньшей мере, 60%, по меньшей мере, 70%, по меньшей мере, 80% или, по меньшей мере, 90% полипептпида, присутствующего в клетке, являются растворимыми.
Полипептиды NIF
В контексте данного документа термины «полипептид Nif» и «белок Nif» используются взаимозаменяемо и означают полипептид, который по аминокислотной последовательности родственен природным полипептидам, участвующим в активности нитрогеназы, где полипептид Nif, согласно изобретению, выбирается из группы, состоящий из: полипептида NifD, полипептида NifH, полипептида NifK, полипептида NifB, полипептида NifE, полипептида NifN, полипептида NifF, полипептида NifJ, полипептида NifM, полипептида NifQ, полипептида NifS, полипептида NifU полипептида NifV, полипептида NifW, полипептида NifX, полипептида NifY и полипептида NifZ, каждый из которых определен в настоящем документе. Полипептиды Nif, согласно изобретению, содержат «слитые полипептиды Nif», которые, в контексте данного документа, означают полипептидный гомолог встречающегося в природе полипептида Nif, который имеет дополнительные аминокислотные остатки, присоединенные к N-концу или C-концу, или к обоим, относительно соответствующего встречающегося в природе полипептида Nif. Как упомянуто выше, слитой полипептид Nif может не иметь инициатора трансляции Met или двух N-концевых остатков Met, относительно соответствующего полипептида Nif дикого типа. Аминокислотные остатки слитого полипептида Nif, которые соответствуют встречающемуся в природе полипептиду Nif, т. е. без дополнительных аминокислотных остатков, присоединенных к N-концу или C-концу, или к обоим, также называются здесь полипептидом Nif, сокращенно «NP» в данном случае, или полипептидом NifD («ND») и т. д. В предпочтительном варианте осуществления «дополнительные аминокислотные остатки, присоединенные к N-концу или C-концу, или к обоим», содержат митохондриальный нацеливающий пептид ( MTP) или процессированный MTP, присоединенный к N-концу NP, или эпитопную последовательность («метка»), которая является N-концевой или C-концевой по отношению к NP или обоим, или одновременно MTP или процессированный MTP и эпитопной последовательности.
Встречающиеся в природе полипептиды Nif встречаются только у некоторых бактерий, включая азотфиксирующие бактерии, включая свободноживущие азотфиксирующие бактерии, ассоциативные азотфиксирующие бактерии и симбиотические азотфиксирующие бактерии. Свободноживущие азотфиксирующие бактерии способны фиксировать значительные уровни азота без прямого взаимодействия с другими организмами. Указанные свободноживущие азотфиксирующие бактерии, не ограничиваясь, содержат представителей видов Azotobacter, Beijerinckia, Klebsiella, Cyanobacteria (классифицируемых как аэробные организмы) и представителей видов Clostridium, Desulfovibrio и названы пурпурными серобактериями, пурпурными несеробактериями и зелеными серобактериями. Ассоциативные азотфиксирующие бактерии представляют собой те прокариотические организмы, которые способны образовывать тесные ассоциации с некоторыми представителями семейства Poaceae (злаковых). Эти бактерии фиксируют заметное количество азота в ризосфере растений-хозяев. Представители видов Azospirillum являются представителями ассоциативных азотфиксирующих бактерий. Симбиотические азотфиксирующие бактерии представляют собой те бактерии, которые симбиотически фиксируют азот, вступая в партнерские отношения с растением-хозяином. Растение вырабатывает сахар в ходе фотосинтеза, который используется азотфиксирующими бактериями для получения энергии, необходимой ему для фиксации азота. Представители видов Rhizobia являются представителями ассоциативных азотфиксирующих бактерий.
Полипептид Nif или слитой полипептид Nif по изобретению выбирются из группы, состоящей из полипептидов: NifH, NifD, NifK, NifB, NifE, NifN, NifF, NifJ, NifM, NifQ, NifS, NifU, NifV, NifW, NifX, NifY и NifZ. Функциональность этих полипептидов была недавно рассмотрена в работе Burén et al. (2020).
Считается, что согласно изобретению, эти полипептиды представляют собой полипептиды VnfG и AnfG, участвующие, соответственно, в V-нитрогеназе и Fe-нитрогеназе в факторах, связанных с нитогеназой (полипептиды Naf), например, NafY, и полипептидах ферредоксина, например, полипептидах FdxN. Эти полипептиды предпочтительно кодируются и экспрессируются как MTP-слитые полипептиды для митохондриального нацеливания.
Полипептид или класс полипептидов можно определить по степени идентичности (% идентичности) его аминокислотной последовательности по отношению к эталонной аминокислотной последовательности и/или по присутствию определенных аминокислотных мотивов или доменов семейства белков, или по наличию большего % идентичности одной эталонной аминокислотной последовательности по сравнению с другой. Полипептид или класс полипептидов также можно определить по тому, обладает ли он такой же биологической активностью, что и природный полипептид Nif, в дополнение к степени идентичности последовательности.
Величину % идентичности полипептида определяют с помощью анализа GAP (Needleman и Wunsch, 1970) (программа GCG) со штрафом за создание GAP=5 и штрафом за продолжение GAP=0,3, или с помощью программы Blastp версии 2.5 или ее обновленных версий (Altschul et al. al., 1997), где в каждом случае анализ выравнивает две последовательности, включая эталонную последовательность, по всей длине эталонной последовательности. В контексте данного документа, эталонные последовательности содержат эти последовательности у встречающихся в природе полипептидов Nif из K. pneumoniae (переименованной в K. oxytoca), SEQ ID NO:1-17.
В следующих определениях степень идентичности аминокислотной последовательности по отношению к эталонной последовательности, представленной как SEQ ID NO, определяется программой Blastp, версии 2.5 или ее обновленными версиями (Altschul et al. 1997), с использованием параметров по умолчанию, за исключением максимального количества целевых последовательностей, которое задано равным 10 000 и определяется по всей длине эталонной аминокислотной последовательности.
Полипептид NifH у встречающихся в природе бактерий является структурным компонентом комплекса нитрогеназы и часто называется железосодержащим (Fe) белком. Он образует гомодимер с кластером Fe4S4, связанным между субъединицами и двумя ATP-связывающими доменами. NifH является облигатным донором электронов по отношению к белку нитрогеназы (гетеротетрамер NifD/NifK) и, следовательно, функционирует как редуктаза нитрогеназы (EC 1.18.6.1). NifH молибденового типа также участвует в биосинтезе FeMo-ко и созревании белка апо-MoFe (Jasniewski et al., 2018). Как указано в этой работе, NifH выполняет три основные функции: (i) участие во встраивании Mo и гомоцитрата в синтез FeMo-ко, также с участием комплекса NifE-NifN, (ii) участие редуктазы в образовании P-кластера в NifD-NifK из того, что называется P * кластер, который также может включать небольшой шапероноподобный полипептид NifZ, и (iii) в участие качестве донора электронов по отношению к белку нитрогеназы.
В контексте данного документа, «полипептид NifH» означает полипептид, содержащий аминокислоты, последовательность которых, по меньшей мере, на 41% идентична аминокислотной последовательности, представленной как SEQ ID NO:1, и которая включает один или несколько доменов TIGR01287, PRK13236, PRK13233 и cd02040. Домен TIGR01287 присутствует в каждой из молибден-железной редуктазы нитрогеназы (NifH), ванадий-железной редуктазы нитрогеназы (VnfH) и железо-железной редуктазы нитрогеназы (AnfH), но исключает гомологичный белок из светонезависимой протохлорофиллидредуктазы. В контексте данного документа, полипептиды NifH содержат подкласс железосвязывающих полипептидов, которые содержат аминокислоты, последовательность которых, по меньшей мере, идентична на 41% SEQ ID NO:1, железосвязывающим полипептидам VnfH и железосвязывающим полипептидам AnfH. Природный полипептид NifH обычно имеет в длину от 260 до 300 аминокислот, а природный мономер имеет молекулярную массу, составляющую примерно 30 кДа. Было идентифицировано большое количество полипептидов NifH, а многочисленные последовательности имеются в общедоступных базах данных. Например, полипептиды NifH, обнаруженные: у Klebsiella michiganensis (Учетный № WP_049123239.1, на 99% идентичны SEQ ID NO:1); у Brenneria goodwinii (WP_048638817.1, идентичны на 93%); Sideroxydans lithotrophicus (WP_013029017.1, идентичны на 84%); у Denitrovibrio acetiphilus (WP_013010353.1, идентичны на 80%); у Desulfovibrio africanus (WP_014258951.1, идентичны на 72%); у Chlorobium phaeobacteroides (WP_011744626.1, идентичны на 69%); у Methanosaeta concilii (WP_013718497.1, идентичны на 64%); у Rhodobacter (WP_009565928.1, идентичны на 61%); у Methanocaldococcus infernus (WP_013099472.1, идентичны на 42%); и у Desulfosporosinus youngiae (WP_007781874.1, идентичны на 41%). Полипептиды NifH описаны и проанализированы в работах Thiel et al. (1997), Pratte et al. (2006), Boison et al. (2006) и Staples et al. (2007).
В контексте данного документа, функциональный полипептид NifH представляет собой полипептид NifH, который способен образовывать функциональный белковый комплекс нитрогеназы вместе с другими необходимыми субъединицами, например, NifD и NifK, и кофактором FeMo, FeV или FeFe.
В данном контексте «полипептид AnfH» представляет собой полипептид NifH, который является элементом консервативного суперсемейства cl25403 (TIGR01287), содержащего консервативный домен PRK13233 и, по меньшей мере, идентичен на 69% аминокислотной последовательности полипептида AnfH Azotobacter vinelandii (SEQ ID NO:218; Учетный № WP_012703362) при измерении полноразмерной SEQ ID NO:218. Эта аминокислотная последовательность используется здесь как эталонная последовательность для AnfH. TIGR01287:AnfH представляет собой полностью железный вариант компонента II нитрогеназы, также известного как редуктаза нитрогеназы. В контексте данного документа, полипептиды AnfH представляют собой подмножество полипептидов NifH. Полипептиды AnfH не включают полипептиды NifH молибденового типа и полипептиды NifH ванадиевого типа (VnfH). Аминокислотные последовательности полипептидов AnfH в базах данных последовательностей обычно аннотируются как полипептиды AnfH. В базе данных белков NCBI по состоянию на январь 2020 года, в наборе AnfH содержалось 314 специфичных аминокислотных последовательностей, каждая из которых имела аминокислотные остатки, специфичные для AnfH и отличающиеся от NifH и VnfH молибденового типа, хотя эти подмножества выглядели более похожими. но все же отличающимися. Примеры встречающихся в природе полипептидов AnfH включают полипептиды AnfH из Rhodocyclus tenuis (Учетный №. WP_153472986; идентичны на 92,36%); Dickeya paradisiaca(Учетный №.WP_015854293; идентичны на 88,36%); Thermodesulfitimonas autotrophica (Учетный №. WP_123927773; идентичны на 78,91%); Clostridium kluyveri (Учетный №.WP_073538802; идентичны на 76,36%); и Methanophagales archaeon (Учетный №. RCV64832; идентичны на 69,37%), каждый со ссылкой на SEQ ID NO:218.
Как описано в Примере 23 данного документа, 16 аминокислот были идентифицированы в определенных позициях в последовательностях AnfH, которые были консервативными и характерными для полипептидов AnfH по сравнению с последовательностью NifH молибденового типа AvNifH. Их можно использовать для отличия полипептидов AnfH от других последовательностей NifH, которые не имеют всех 16 общих аминокислот. AvNifH, KoNifH (SEQ ID NO: 1) и другие последовательности NifH молибденового типа имели мотивы III и IV, но не имели мотивов I, II, V-VII, и поэтому эти мотивы (SEQ ID NO:225-231) также могли использоваться для отличия подмножества AnfH от других полипептидов NifH.
Аналогично другим функциональным полипептидам NifH, функциональные полипептиды AnfH способны функционировать как редуктаза нитрогеназы, являясь облигатным донором электронов для комплекса FeFe. Подобно NifH молибденового типа, AnfH потенциально участвует в биосинтезе FeFe-ко и созревании комплекса апо-FeFe (AnfD-AnfK-AnfG).
В данном контексте «полипептид NifD» означает полипептид, содержащий аминокислоты, последовательность которых, по меньшей мере, идентична на 33% аминокислотной последовательности, представленной как SEQ ID NO:2, и который включает: (i) один или оба домена TIGR01282 и COG2710, оба из которых обнаружены в полипептидах, связывающих железо-молибден, включая полипептид, имеющий аминокислотную последовательность, показанную в SEQ ID NO:2; или (ii) железо-ванадиевый связывающий домен TIGR01860, когда полипептид NifD находится в подклассе полипептидов VnfD; или (iii) железо-железосвязывающий домен TIGR1861, когда полипептид NifD находится в подклассе полипептидов AnfD. Полипептид NifD может представлять собой часть слитого полипептида, например, слитого с MTP и/или NifK, или, альтернативно, может не содержать каких-либо N- или C-концевых продолжений. В предпочтительном варианте осуществления, полипептид NifD, связанный с полипептидом NifK, связывает FeMo-кофактор.
В контексте данного документа, полипептиды NifD включают подкласс полипептидов, связывающих железо-молибден (FeMo-ко), содержащих аминокислоты, последовательность которых, по меньшей мере, идентична на 33% SEQ ID NO:2, железо-ванадиевым полипептидам VnfD и полипептидам AnfD. Природный полипептид NifD обычно имеет в длину от 470 до 540 аминокислот. Было идентифицировано большое количество полипептидов NifD а многочисленные последовательности имеются в общедоступных базах данных. Например, полипептиды NifH, обнаруженные: у Raoultella ornithinolytica (Учетный №. WP_044347161.1, идентичны на 96% SEQ ID NO:2); у Kluyvera intermedia (WP_047370273.1, идентичны на 93%); у Dickeya dadantii (WP_038902190.1, идентичны на 89% ); у Tolumonas sp. BRL6-1 (WP_024872642.1, идентичны на 81%); у Magnetospirillum gryphiswaldense (WP_024078601.1, идентичны на 68%); у Thermoanaerobacterium thermosaccharolyticum (WP_013298320.1, идентичны на 42%); у Methanothermobacter thermautotrophicus (WP_010877172.1, идентичны на 38%); у Desulfovibrio africanus (WP_014258953.1, идентичны на 37%); у Desulfotomaculum sp. LMa1 (WP_066665786.1, идентичны на 37%); у Desulfomicrobium baculatum (WP_015773055.1, идентичны на 36%); у полипептида VnfD из Fischerella muscicola (WP_016867598.1, идентичны на 34%); а у полипептида AnfD из Opitutaceae bacterium TAV5 (WP_009512873.1, идентичны на 33%). Полипептиды NifD были описаны и проанализированы в работах Lawson и Smith (2002), Kim и Rees (1994), Eady (1996), Robson et al. (1989), Dilworth et al. (1988), Dilworth et al. (1993), Miller и Eady (1988), Chiu et al. (2001), Mayer et al. (1999), и Tezcan et al. (2005).
Полипептиды NifD железо-молибденового подкласса являются главной субъединицей комплексов нитрогеназы, являясь α субъединицей белкового комплекса α2β2 MoFe в ядре нитрогеназы и местом восстановления субстрата FeMo-кофактором. В контексте данного документа, функциональный полипептид NifD представляет собой полипептид NifD который способен образовывать функциональный белковый комплекс нитрогеназы вместе с другими необходимыми субъединицами, например, NifH и NifK, и FeMo или прочими кофакторами.
В контексте данного документа, термин «полипептид NifD (ND), устойчивый к расщеплению протеазой», устойчив к расщеплению в определенном сайте или в определенной области, например, в аминокислотной последовательности, соответствующей аминокислотам 97-100 из SEQ ID NO:18, когда ND вводится в митохондрии растений с помощью MTP. В контексте данного документа, термин «устойчивый к расщеплению протеазой» означает получение <10% расщепления, когда полипептид NifD вводится в митохондрии растений с использованием MTP. В предпочтительных вариантах осуществления менее 5% полипептида NifD расщепляется на сайте или внутри области, более предпочтительно по существу не расщепляется, или расщепление не обнаруживается. Полипептид NifD может быть «относительно устойчивым к расщеплению» по сравнению с полипептидом NifD, содержащим аминокислотную последовательность, представленную как SEQ ID NO:18, который расщепляется, по меньшей мере, в 5 раз реже, предпочтительно по меньшей мере в 10 раз реже, по сравнению с полипептидом NifD, содержащим аминокислотную последовательность, представленную как SEQ ID NO:18.
В контексте данного документа, «аминокислотная последовательность, отличная от RRNY (SEQ ID NO:101) в позициях, соответствующих аминокислотам 97-100 из SEQ ID NO:18», относится к последовательности, которая включает четыре остатка в позициях, соответствующих аминокислотам 97-100 из SEQ ID NO:18, которая не является RRNY.
В контексте данного документа, «полипептид AnfD» представляет собой полипептид NifD, который является специфичным элементом консервативного суперсемейства cl30843 оксидоредуктазы нитрогеназы, содержащего консервативный домен TIGR01861 и, по меньшей мере, идентичен на 71% аминокислотной последовательности полипептида AnfD Azotobacter vinelandii (SEQ ID NO: 216; Учетный № WP_012703361) при измерении полноразмерной SEQ ID NO: 216. Эта аминокислотная последовательность используется здесь как эталонная последовательность для AnfD. TIGR01861:AnfD представляет собой полностью железный вариант компонента I α-цепи нитрогеназы. В контексте данного документа, полипептиды AnfD представляют собой подмножество полипептидов NifD. Полипептиды AnfD не содержат полипептиды NifD молибденового типа и полипептиды NifD ванадиевого типа (VnfD), а также не содержат полипептиды протохлорофиллид- или хлорофиллид-редуктазы (Boyd и Peters, 2013). Аминокислотные последовательности полипептидов AnfD в базах данных белковых последовательностей обычно аннотируются как полипептиды AnfD. По состоянию на январь 2020 года, в наборе AnfD базы данных белков NCBI было 156 специфичных аминокислотных последовательностей. Примеры встречающихся в природе полипептидов AnfH включают полипептиды AnfD из Desulfovibrio sp. DV (Учетный №. WP_075356167; идентичны на 87,47%); Paenibacillus sp. FSL H7-0357 (Учетный №. WP_038590013; идентичны на 85,52%); Rhodobacter capsulatus (Учетный №. WP_ 023922817; идентичны на 80,31%); Methanosarcina acetivorans C2A (Учетный №. WP_011021232; идентичны на 77,13%); и Bacteroidales bacterium Barb7 (Учетный №. OAV73823; идентичны на 71,25%), каждый со ссылкой на SEQ ID NO:216. Дополнительные примеры приведены в работе McRose et al. (2017).
Аналогично другим полипептидам NifD, которые являются функциональными, функциональные полипептиды AnfD способны функционировать в качестве структурного компонента α-белка гетерогексамерной нитрогеназы α2β2δ2 с β-белком (AnfK) и δ-белком (AnfG), обеспечивая связывание каталитического комплекса FeFe-ко для снижения молекулярного азота.
В контексте данного документа, «полипептид NifK» означает полипептид, содержащий аминокислоты, последовательность которых, по меньшей мере, идентична на 31% аминокислотной последовательности, представленной как SEQ ID NO:3, и который включает один или несколько консервативных доменов cd01974, TIGR01286 или cd01973, когда полипептид NifK относится к подклассу полипептидов VnfK или cl02775, содержащему консервативный домен TIGR02931, когда полипептид NifK находится в подклассе полипептидов AnfK. В контексте данного документа, полипептиды NifK содержат полипептиды VnfK из железо-ванадиевой нитрогеназы и железосвязывающие полипептиды AnfK. Природный полипептид NifK обычно имеет в длину от 430 до 530 аминокислот. Было идентифицировано большое количество полипептидов NifK, а многочисленные последовательности имеются в общедоступных базах данных. Например, полипептиды NifK обнаруженные: у Klebsiella michiganensis (Учетный № . WP_049080161.1, идентичны на 99% SEQ ID NO:3); у Raoultella ornithinolytica (WP_044347163.1, идентичны на 96%); у Klebsiella variicola (SBM87811.1, идентичны на 94%); у Kluyvera intermedia (WP_047370272.1, идентичны на 89%); у Rahnella aquatilis (WP_014333919.1, идентичны на 82%); у Tolumonas auensis (WP_012728880.1, идентичны на 75%); у Pseudomonas stutzeri (WP_011912506.1, идентичны на 68%); у Vibrio natriegens (WP_065303473.1, идентичны на 65%); у Azoarcus toluclasticus (WP_018989051.1, идентичны на 54%); у Frankia sp. (prf||2106319A, идентичны на 50%) и у Methanosarcina acetivorans (WP_011021239.1, идентичны на 31%). В базах данных, помеченных как «NifK», есть несколько примеров полипептидов, которые идентичны менее чем на 31% с SEQ ID NO:3, но не содержат ни одного из доменов, перечисленных выше, и поэтому не включены в настоящее описание в качестве полипептидов NifK. Полипептиды NifK были описаны и проанализированы в работах Kim и Rees (1994), Eady (1996), Robson et al. (1989), Dilworth et al. (1988), Dilworth et al. (1993), Miller и Eady (1988), Igarashi и Seefeldt (2003), Fani et al. (2000) и Rubio и Ludden (2008).
Полипептиды NifK железо-молибденового подкласса являются главной субъединицей комплексов нитрогеназы, являясь β субъединицей белкового комплекса α2β2 MoFe в ядре нитрогеназы. В контексте данного документа, функциональный полипептид NifK представляет собой полипептид NifK который способен образовывать функциональный белковый комплекс нитрогеназы вместе с другими необходимыми субъединицами, например, NifD и NifH, и FeMo или прочими кофакторами. В предпочтительном варианте осуществления, при выравнивании с аминокислотной последовательностью SEQ ID NO:3, аминокислотная последовательность полипептида NifK, согласно изобретению, имеет на своем С-конце аминокислоты DLVR (SEQ ID NO:58), причем аргинин представляет собой С-концевую аминокислоту. То есть полипептид NifK и слитой полипептид NifK, согласно изобретению, предпочтительно имеет тот же С-конец, что и нативные полипептиды NifK, то есть он не имеет искусственного добавления к С-концу. Такие предпочтительные полипептиды NifK лучше способны образовывать функциональный нитрогеназный комплекс с полипептидами NifD и NifH.
Полипептиды NifK железо-молибденового подкласса являются главной субъединицей комплексов нитрогеназы, являясь β субъединицей белкового комплекса α2β2 MoFe в ядре нитрогеназы. В контексте данного документа, функциональный полипептид NifK представляет собой полипептид NifK который способен образовывать функциональный белковый комплекс нитрогеназы вместе с другими необходимыми субъединицами, например, NifD и NifH, и FeMo или прочими кофакторами. В предпочтительном варианте осуществления, при выравнивании с аминокислотной последовательностью SEQ ID NO:3, аминокислотная последовательность слитого полипептида NifK и расщепленный полипептид NifK, согласно изобретению, имеет на своем С-конце аминокислоты DLVR (SEQ ID NO:58), причем аргинин представляет собой С-концевую аминокислоту. В других предпочтительных вариантах осуществления, аминокислотная последовательность слитого полипептида NifK и расщепленного полипептида NifK, согласно изобретению, имеет на своем С-конце аминокислотную последовательность DLIR (SEQ ID NO:239), DVVR (SEQ ID NO:240), DIIR (SEQ ID NO:241), DLTR (SEQ ID NO:242) или INVW (SEQ ID NO:243), которые обычно не присутствуют в нативных последовательностях AnfK. Полипептид NifK и слитой полипептид NifK, согласно изобретению, и отщепленный от него полипептид NifK предпочтительно имеет тот же С-конец, что и нативный полипептид NifK, т. е. он не имеет искусственного добавления к С-концу, и не имеет никаких аминокислот, удаленных с С-конца при выравнивании с помощью нативного полипептида NifK. Такие предпочтительные полипептиды NifK лучше способны образовывать функциональный нитрогеназный комплекс с полипептидами NifD и NifH.
В контексте данного документа, «полипептид AnfK» представляет собой полипептид, который является специфичным элементом консервативного суперсемейства cl02775 оксидоредуктазы нитрогеназы, содержащего консервативный домен TIGR02931 и, по меньшей мере, идентичен на 54% аминокислотной последовательности полипептида AnfD Azotobacter vinelandii (SEQ ID NO:217; Учетный № WP_012703359) при измерении полноразмерной SEQ ID NO:217. Эта аминокислотная последовательность используется здесь как эталонная последовательность для AnfK. TIGR02931:AnfK представляет собой полностью железный вариант компонента I β-цепи нитрогеназы. В контексте данного документа, полипептид AnfK может представлять собой полипептид NifK, который, по меньшей мере, идентичен на 31% аминокислоте с SEQ ID NO:3. Другие полипептиды AnfK менее гомологичны и идентичны только на 25-31% SEQ ID NO:3, но, тем не менее, включены в полипептиды AnfK, согласно изобретению. Полипептиды AnfK не включают полипептиды NifK молибденового типа и полипептиды NifK ванадиевого типа (VnfK). Слитой полипептид AnfK и расщепленный полипептид AnfK, согласно изобретению, предпочтительно имеют тот же С-конец, что и нативный полипептид AnfK, т. е. он не имеет искусственного добавления к С-концу и не имеет никаких аминокислот, удаленных из С-конца при выравнивании с нативным полипептидом AnfK, например SEQ ID NO:217. В предпочтительных вариантах осуществления, аминокислотная последовательность слитого полипептида AnfK и расщепленного полипептида AnfK имеет на своем С-конце аминокислотные последовательности LNVW (SEQ ID NO:244), LNTW (SEQ ID NO:245), LNMW (SEQ ID NO:246), LAMW (SEQ ID NO:247) или LSVW (SEQ ID NO:248). Аминокислотные последовательности полипептидов AnfK в базах данных белковых последовательностей обычно аннотируются как полипептиды AnfK. В базе данных белков NCBI по состоянию на январь 2020 года, в наборе AnfH содержалось 155 специфичных аминокислотных последовательностей, каждая из которых отличалось от NifK молибденового типа и полипептидных последовательностей VnfK. Примеры встречающихся в природе полипептидов AnfK включают полипептиды AnfK из Azomonas agilis (Учетный №. WP_144571040; идентичны на 91,34%), Clostridium sp. BL-8 (Учетный №. WP_077859050; идентичны на 78,35%), Lucifera butyrica (Учетный №. WP_122630336; идентичны на 62,34%) и Rhodoblastus acidophilus (Учетный №. WP_088520366; идентичны на 54%), каждый со ссылкой на SEQ ID NO:217.
Аналогично другим полипептидам NifK которые являются функциональными, функциональные полипептиды AnfK способны функционировать в качестве структурного компонента β белка гетерогексамерной нитрогеназы α2β2δ2 с α белком (AnfD) и δ-белком (AnfG), обеспечивая формирование комплекса, имеющего активный сайт для снижения молекулярного азота в FeFe-ко.
Полипептид NifB в природных бактериях представляет собой белок, который преобразует кластеры [4Fe-4S] в NifB-ко, кластер Fe-S с более высокой нуклеарностью с центральным атомом C, который служит предшественником FeMo-ко, FeV-ко и синтезу FeFe-ко (Guo et al., 2016). Таким образом, NifB катализирует первую коммитированную стадию в путях синтеза FeMo-ко, FeV-ко и FeFe-ко и, следовательно, важен для функциональности нитрогеназы. NifB-ко, который является продуктом NifB способен связываться с комплексом NifE-NifN и может перемещаться от NifB к NifE-NifN с помощью белка-носителя NifX металлокластера.
В контексте данного документа, «полипептид NifB» означает полипептид, аминокислотная последовательность которого включает аминокислоты, последовательность которых, по меньшей мере, идентична на 27% аминокислотной последовательности, представленной как SEQ ID NO:4. Большинство полипептидов NifB содержат один или несколько консервативных домена TIGR01290, консервативных домена NifB cd00852, консервативных домена суперсемейства NifX-NifB cl00252 и консервативных домена Radical_SAM cd01335. В контексте данного документа, полипептиды NifB одержат встречающиеся в природе полипептиды, которые были аннотированы как имеющие функциональность NifB, но не имеющие ни одного из этих доменов. Полипептиды NifB из Klebsiella, Azotobacter, Rhizobium, Bradyrhizobium и других бактерий имеют С-концевое NifX-подобное продолжение, тогда как большинство архейных полипептидов NifB лишены NifX-подобного домена и называются «укороченными полипептидами NifB». Природный полипептид NifB обычно имеет в длину от 440 до 500 аминокислот, а природный мономер имеет молекулярную массу, составляющую примерно 50 кДа. Было идентифицировано большое количество полипептидов NifB, а многочисленные последовательности имеются в общедоступных базах данных. Например, полипептиды NifB, обнаруженные: у Raoultella ornithinolytica (Учетный №. WP_041145602.1, идентичны на 91% SEQ ID NO:4); у Kosakonia radicincitans (WP_043953592.1, идентичны на 80%); у Dickeya chrysanthemi (WP_040003311.1, идентичны на 76%); у Pectobacterium atrosepticum (WP_011094468.1, идентичны на 70%); у Brenneria goodwinii (WP_048638849.1, идентичны на 63%); у Halorhodospira halophila (WP_011813098.1, идентичны на 59%, без домена NifX); у Methanosarcina barkeri (WP_048108879.1, идентичны на 50%, без домена NifX); у Clostridium purinilyticum (WP_050355163.1, идентичны на 40% без домена NifX) и у Desulfovibrio salexigens (WP_015850328.1, идентичны на 27%). В контексте данного документа, «функциональный полипептид NifB» представляет собой полипептид NifB, который способен образовывать NifB-ко из кластеров [4Fe-4S]. Функциональный NifB требует использования S-аденозилметионина (SAM) для осуществления своей функции. Полипептиды NifB были описаны и проанализированы в работах Curatti et al. (2006) и Allen et al. (1995).
Boyd et al. (2011) исследовали филогенетические отношения Anf/Vnf/NifDKEN и NifB из 40 таксонов и сделали следующие выводы: (1) Горизонтальный перенос генов кластера Nif, кодирующего NifB, лишенного C-концевого домена NifX, произошел от предка метаногена в порядке Methanosarcinales к анаэробному предку Firmicutes, где два организма сосуществовали в анаэробной среде и где был доступен молибден, и (2) после этого события горизонтального переноса генов произошло слияние NifB и NifX в Firmicutes, от которого произошла диазотрофная бактериальная клеточна линия. В поддержку этой теории были представлены следующие доказательства: (1) Ни одна из метаногенных архей (Methanococcales, Methanosarcinales и Methanobacteriales) не имеет NifB с С-концевым доменом NifX, (2) последовательности NifB из Methanobacteriales и Methanococcales указывают на раннюю дивергенцию из последовательностей Methanosarcinales и Bacteria, и (3) некоторые из анаэробных Firmicutes, Chloroflexi и Proteobacteria, которые имеют NifB без C-концевого домена NifX, рано отошли от клеточной линии Firmicute, предположительно вскоре после события горизонтального переноса генов Nif.
Чтобы определить наличие или отсутствие C-концевого домена NifX в полипептидах NifB, аминокислотную последовательность NifB можно выровнять с помощью инструмента множественного выравнивания на основе ограничений (COBALT, NCBI, www.ncbi.nlm.nih.gov/tools/cobalt/re_cobalt.cgi) с репрезентативными последовательностями NifB, такими как Klebsiella michiganensis NifB (Учетный № P10930), Klebsiella michiganensis NifX (KZT46636.1), NifY (KZT46633.1), A. vinelandii NifX (AGK13791.1), NifY ( AGK13792.1), NafY (AGK13761.1) и белок семейства NifX/NifY/NafY/VnfX (AGK14217.1). «Сайт связывания FeMo-кофактора динитрогеназы» (семейство Pfam PF02579) в каждой последовательности можно идентифицировать с помощью PfamScan (EMBL-EBI, www.ebi.ac.uk/Tools/pfa/pfamscan/), используя базу данных Pfam-A со значением ожидания, установленным на 10.
Комплекс NifEN представляет собой каркасный комплекс, необходимый для правильной сборки динитрогеназы, функционирующий как каркас для созревания NifB-ко в FeMo-ко, для которого также требуется функциональность NifH, а также он структурно подобен динитрогеназе (Fay et al. , 2016). Комплекс NifEN состоит из 2 субъединиц каждого из NifE и NifN, соответственно, образующих гетеротетрамер, обозначаемый здесь ENα2β2. Полипептид NifE в природных бактериях представляет собой полипептид, который является α субъединицей тетрамера ENα2β2 с полипептидом NifN, и этот тетрамер ENα2β2 необходим для синтеза FeMo-ко и, как предполагается, действует как каркас, на котором синтезируется FeMo-ко.
В контексте данного документа, «полипептид NifE» означает полипептид, содержащий аминокислоты, последовательность которых, по меньшей мере, на 32% идентична аминокислотной последовательности, представленной как SEQ ID NO:5, и которая включает один или несколько доменов TIGR01283 и PRK14478. Элементы семейства белков домена TIGR01283 также являются элементами суперсемейства cl02775. Природный полипептид NifE обычно имеет в длину от 440 до 490 аминокислот, а природный мономер имеет молекулярную массу, составляющую примерно 50 кДа. Было идентифицировано большое количество полипептидов NifE, а многочисленные последовательности имеются в общедоступных базах данных. Например, полипептиды NifE обнаруженные: у Klebsiella michiganensis (Учетный №. WP_049114606.1, идентичны на 99% SEQ ID NO:5); у Klebsiella variicola (SBM87755.1, идентичны на 92%); у Dickeya paradisiaca (WP_012764127.1, идентичны на 89%); у Tolumonas auensis (WP_012728883.1, идентичны на 75%); у Pseudomonas stutzeri (WP_003297989.1, идентичны на 69%); у Azotobacter vinelandii (WP_012698965.1, идентичны на 62%); у Trichormus azollae (WP_013190624.1, идентичны на 55%); у Paenibacillus durus (WP_025698318.1, идентичны на 50%); у Sulfuricurvum kujiense (WP_013460149.1, идентичны на 44%): у Methanobacterium formicicum (AIS31022.1, идентичны на 39%); у Anaeromusa acidaminophila (WP_018701501.1, идентичны на 35%) и у Megasphaera cerevisiae (WP_048514099.1, идентичны на 32%). В контексте данного документа, термин «функциональный полипептид NifE» представляет собой полипептид NifE, который способен образовывать функциональный тетрамер вместе с NifN таким образом, что данный комплекс способен синтезировать FeMo-ко. Этот синтез FeMo-ко включает другие полипептиды, включая NifH и NifB, и может включать NifX. Полипептиды NifE описаны и проанализированы в работах Fay et al. (2016), Hu et al. (2005), Hu et al. (2006) и Hu et al. (2008).
Полипептид NifF у встречающихся в природе диазотрофов представляет собой флаводоксин, который является донором электронов для NifH. В контексте данного документа «полипептид NifF» означает полипептид, содержащий аминокислоты, последовательность которых, по меньшей мере, на 34% идентична аминокислотной последовательности, представленной как SEQ ID NO:6, и которая включает один или оба долгоживущих домена флаводоксина, домена TIGR01752 и домена FLDA флаводоксина, обнаруженного в белках Nif из Azobacter и другого бактериального вида PRK09267. Полипептиды NifF включают флаводоксины, связанные с активацией пируватформиатлиазы и кобаламин-зависимой активностью метионинсинтазы у нефиксирующих азот бактерий, но исключают другие флаводоксины, участвующие в более широких функциях. Природный полипептид NifF обычно имеет в длину от 160 до 200 аминокислот, а природный мономер имеет молекулярную массу, составляющую примерно 19 кДа. Было идентифицировано большое количество полипептидов NifF а многочисленные последовательности имеются в общедоступных базах данных. Например, полипептиды NifF, обнаруженные: у Klebsiella michiganensis (Учетный №. WP_004122417.1, идентичны на 99% SEQ ID NO:6); у Klebsiella variicola (WP_040968713.1, идентичны на 85%); у Kosakonia radicincitans (WP_035885760.1, идентичны на 76%); у Dickeya chrysanthemi (WP_039999438.1, идентичны на 72%); у Brenneria goodwinii (WP_048638838.1, идентичны на 62%); у Methylomonas methanica (WP_064006977.1, идентичны на 56%); у Azotobacter vinelandii (WP_012698862.1, идентичны на 50%); у Chlorobaculum tepidum (WP_010933399.1, идентичны на 39%); у Campylobacter showae (WP_002949173.1, идентичны на 37%) и у Azotobacter chromococcum (WP_039801725.1, идентичны на 34%). В контексте данного документа, «функциональный полипептид NifF» представляет собой полипептид NifF который способен быть донором электронов для полипептида NifH. Полипептиды NifX были описаны и проанализированы в работах Drummond (1985).
В контексте данного документа, «полипептид AnfG» является элементом консервативного суперсемейства cl03910 нитрогеназы (pfam03139-AnfG), содержащего консервативный домен TIGR02929 и, по меньшей мере, идентичен на 42% аминокислотной последовательности полипептида AnfD Azotobacter vinelandii (SEQ ID NO:219; Учетный № WP_012703360) при измерении полноразмерной SEQ ID NO:219. Эта аминокислотная последовательность используется здесь как эталонная последовательность для AnfG. TIGR02929 представляет собой полностью железный вариант компонента I δ-цепи нитрогеназы. Полипептиды AnfG не включают полипептиды NifG ванадиевого типа (VnfG). Аминокислотные последовательности полипептидов AnfG в базах данных белковых последовательностей обычно аннотируются как полипептиды AnfG. По состоянию на январь 2020 года, в наборе AnfG базы данных белков было 150 специфичных аминокислотных последовательностей. Примеры встречающихся в природе полипептидов AnfG включают полипептиды AnfG из Azomonas agilis (Учетный №. WP_144571041; идентичны на 84,73%), Firmicutes bacterium (Учетный №. HBE76208; идентичны на 70,37%), Sporomusa termitida (Учетный №. WP_144349445; идентичны на 68,75%), Rhodovulum viride (Учетный №. WP_112317428; идентичны на 57,14%) и Megasphaera cerevisiae (Учетный №. WP_048515315; идентичны на 42,86%), каждый со ссылкой на SEQ ID NO:219.
Функциональные полипептиды AnfG способны функционировать в качестве структурного компонента δ-белка гетерогексамерной нитрогеназы α2β2δ2.
Полипептид NifJ в природных бактериях представляет собой пируват:флаводоксин (ферредоксин) оксидоредуктазу, которая является донором электронов для NifH. В контексте данного документа, «полипептид NifJ» означает полипептид, содержащий аминокислоты, последовательность которых, по меньшей мере, на 40% идентична аминокислотной аминокислотного партнера последовательности, представленной как SEQ ID NO:7, и которая включает консервативный домен TIGR02176. Природный полипептид NifJ обычно имеет в длину от 1100 до 1200 аминокислот, а природный мономер имеет молекулярную массу, составляющую примерно 128 кДа. Было идентифицировано большое количество полипептидов NifJ, а многочисленные последовательности имеются в общедоступных базах данных. Например, полипептиды NifJ , обнаруженные: у Klebsiella michiganensis (Учетный №. WP_024360006.1, идентичны на 99% SEQ ID NO:7); у Raoultella ornithinolytica (WP_044347157.1, идентичны на 95%); у Klebsiella quasipneumoniae (WP_050533844.1, идентичны на 92%); у Kosakonia oryzae (WP_064566543.1, идентичны на 82%); у Dickeya solani (WP_057084649.1, идентичны на 78%); у Rahnella aquatilis (WP_014683040.1, идентичны на 72%); у Thermoanaerobacter mathranii (WP_013149847.1, идентичны на 64%); у Clostridium botulinum (WP_053341220.1, идентичны на 60%); у Spirochaeta africana (WP_014454638.1, идентичны на 52%) и у Vibrio cholerae (CSA83023.1, идентичны на 40%). В контексте данного документа, «функциональный полипептид NifJ » представляет собой полипептид NifJ, который способен быть донором электронов для полипептида NifJ. Полипептиды NifJ были описаны и проанализированы в работах Schmitz et al. (2001).
Полипептид NifM в природных бактериях представляет собой полипептид, необходимый для созревания некоторых, но не всех полипептидов NifH. В отсутствие NifM, NifH K. oxytoca присутствовал только на низких уровнях в E. coli и дрожжах при гетерологичной экспрессии и не был способен отдавать электроны для NifD-NifK. В контексте данного документа, «полипептид NifM» означает полипептид, содержащий аминокислоты, последовательность которых, по меньшей мере, идентична на 26% аминокислотной последовательности, представленной как SEQ ID NO:8, и которая включает домен TIGR02933. Полипептиды NifM гомологичны пептидил-пролил-цис-транс-изомеразам (PPIase), группе ферментов, которые способствуют укладыванию белка, катализируя цис-транс-изомеризацию пролиновых имидных пептидных связей, имеющих домен типа PpiC, и, по-видимому, являются вспомогательными белками для некоторых полипептидов NifH, включая, по крайней мере, некоторые полипептиды VnfH и AnfH. Природный полипептид NifM имеет в длину от 240 до 300 аминокислот, а природный мономер имеет молекулярную массу, составляющую примерно 30 кДа. Было идентифицировано большое количество полипептидов NifM а многочисленные последовательности имеются в общедоступных базах данных. Например, полипептиды NifM, обнаруженные: у Klebsiella oxytoca (Учетный №. WP_064342940.1, идентичны на 99% SEQ ID NO:8); у Klebsiella michiganensis (WP_004122413.1, идентичны на 97%); у Raoultella ornithinolytica (WP_044347181.1, идентичны на 85%); у Klebsiella variicola (WP_063105800.1, идентичны на 75%); у Kosakonia radicincitans (WP_035885759.1, идентичны на 59% ); у Pectobacterium atrosepticum (WP_011094472.1, идентичны на 42%); у Brenneria goodwinii (WP_048638837.1, идентичны на 33%); у Pseudomonas aeruginosa PAO1 (CAA75544.1, идентичны на 28%); у Marinobacterium sp. AK27 (WP_051692859.1, идентичны на 27%) и у Teredinibacter turnerae (WP_018415157.1, идентичны на 26%). В контексте данного документа, «функциональный полипептид NifM» представляет собой полипептид NifM который способен образовывать комплекс с полипептидом NifH для созревания полипептида NifH. Полипептиды NifM были описаны и проанализированы в работах Petrova et al. (2000).
Полипептид NifN в природных бактериях представляет собой β субъединицу тетрамера ENα2β2 с полипептидом NifN, и этот тетрамер ENα2β2 необходим для синтеза FeMo-ко и, как предполагается, действует как каркас, на котором синтезируется FeMo-ко. В контексте данного документа, «полипептид NifN» означает (i) полипептид, содержащий аминокислоты, последовательность которых, по меньшей мере, идентична на 76% последовательности, представленной как SEQ ID NO:9, и/или (ii) полипептид, содержащий аминокислоты, последовательность которых, по меньшей мере, идентична на 34% последовательности, представленной как SEQ ID NO:9, которая включает один или несколько консервативных доменов TIGR01285, cd01966 и PRK14476. NifN родственен по структуре β-цепи молибден-железосодержащего белка NifK. Полипептиды, содержащие консервативный TIGR01285, охватывают большинство примеров полипептидов NifN, но исключают некоторые полипептиды NifN, например предполагаемый NifN Chlorobium tepidum, и поэтому определение NifN не ограничивается полипептидами, содержащими консервативный домен TIGR01285. Элементы семейства белков домена PRK14476 также являются элементами суперсемейства cl02775. Природный полипептид NifN обычно имеет в длину от 410 до 470 аминокислот, хотя при естественном слиянии с NifE он может иметь около 900 аминокислотных остатков, а природный мономер имеет молекулярную массу, составляющую примерно 50 кДа. Было идентифицировано большое количество полипептидов NifN многочисленные последовательности имеются в общедоступных базах данных. Например, полипептиды NifN, обнаруженные: у Klebsiella oxytoca (Учетный №. WP_064391778.1, идентичны на 97% SEQ ID NO:9); у Kluyvera intermedia (WP_047370268.1, идентичны на 80%); у Rahnella aquatilis (WP_014683026.1, идентичны на 70%); у Brenneria goodwinii (WP_048638830.1, идентичны на 65%); у Methylobacter tundripaludum (WP_027147663.1, идентичны на 46%); у Calothrix parietina (WP_015195966.1, идентичны на 41%); у Zymomonas mobilis (WP_023593609.1, идентичны на 37%); у Paenibacillus massiliensis (WP_025677480.1, идентичны на 35%) и у Desulfitobacterium hafniense (WP_018306265.1, идентичны на 34%). В контексте данного документа, термин «функциональный полипептид NifN» представляет собой полипептид NifN , который способен образовывать функциональный тетрамер вместе с NifE таким образом, что данный комплекс способен синтезировать FeMo-ко. Полипептиды NifN описаны и рассмотрены в работах Fay et al. (2016), Brigle et al. (1987), Fani et al. (2000), и Hu et al. (2005).
Полипептид NifQ в природных бактериях представляет собой полипептид, участвующий в синтезе FeMo-ко, вероятно, в раннем процессинге MoO42-. Консервативные C-концевые цистеиновые остатки могут участвовать в связывании металлов. В контексте данного документа, «полипептид NifQ» означает полипептид, содержащий аминокислоты, последовательность которых, по меньшей мере, идентична на 34% аминокислотной последовательности, представленной как SEQ ID NO:10, и которая является элементом семейства белков домена CL04826 и элементом семейства белков домена pfam04891. Природный полипептид NifQ обычно имеет в длину от 160 до 250 аминокислот, хотя он может иметь в длину 350 аминокислотных остатков, а природный мономер имеет молекулярную массу, составляющую примерно 20 кДа. Было идентифицировано большое количество полипептидов NifQ последовательности имеются в общедоступных базах данных. Например, полипептиды NifQ, обнаруженные: у Klebsiella oxytoca (Учетный №. WP_064391765.1, идентичны на 95% SEQ ID NO:10); у Klebsiella variicola (CTQ06350.1, идентичны на 75%); у Kluyvera intermedia (WP_047370257.1, идентичны на 63%); у Pectobacterium atrosepticum (WP_043878077.1, идентичны на 59%); у Mesorhizobium metallidurans (WP_008878174.1, идентичны на 46%); у Rhodopseudomonas palustris (WP_011501504.1, идентичны на 42%); у Paraburkholderia sprentiae (WP_027196569.1, идентичны на 41%); у Burkholderia stabilis (GAU06296.1, идентичны на 39%) и у Cupriavidus oxalaticus (WP_063239464.1, идентичны на 34%). В контексте данного документа, «функциональный полипептид NifQ» представляет собой полипептид NifQ, который способен процессировать MoO42-. Полипептиды NifQ были описаны и проанализированы в работах Allen et al. (1995) и Siddavattam et al. (1993).
Полипептид NifS в природных бактериях представляет собой цистеин-десульфуразу, участвующую в биосинтезе железо-серного (FeS) кластера, напр., который участвует в мобилизации серы для синтеза и восстановления кластера Fe-S. В контексте данного документа, «полипептид NifS» означает (i) полипептид, содержащий аминокислоты, последовательность которых, по меньшей мере, идентична на 90% последовательности, представленной как SEQ ID NO:19, и/или (ii) полипептид, содержащий аминокислоты, последовательность которых, по меньшей мере, идентична на 36% последовательности, представленной как SEQ ID NO:19, которая включает один или оба консервативных домена TIGR03402 и COG1104. Семейство белков домена TIGR03402 включает кладу, почти всегда обнаруживаемую в расширенных системах фиксации азота, плюс вторую кладу, более тесно связанную с первой, чем с IscS, а также часть NifS-подобных/NifU-подобных систем. Семейство белков домена TIGR03402 не распространяется на более отдаленную кладу, обнаруженную в протеобактериях epsilon, таких как Helicobacter pylori, также называемая в литературе NifS, построенная вместо этого в TIGR03403. Семейство белков домена COG1104 включает цистеинсульфинатдесульфиназу/цистеиндесульфуразу или родственные ферменты. Некоторые полипептиды NifS включают отдельный домен аминотрансферазы cl18945. Природный полипептид NifS имеет в длину от 370 до 440 аминокислот, а природный мономер имеет молекулярную массу, составляющую примерно 43 кДа. Было идентифицировано большое количество полипептидов NifS, а многочисленные последовательности имеются в общедоступных базах данных. Например, полипептиды NifH, обнаруженные: у Klebsiella michiganensis (Учетный № WP_004138780.1, на 99% идентичны SEQ ID NO:19); у Raoultella terrigena (WP_045858151.1, идентичны на 89%); у Kluyvera intermedia (WP_047370265.1, идентичны на 80%); у Rahnella aquatilis (WP_014333911.1, идентичны на 73%); у Agarivorans gilvus (WP_055731597.1, идентичны на 64%); у Azospirillum brasilense (WP_014239770.1, идентичны на 60%); у Desulfosarcina cetonica (WP_054691765.1, идентичны на 55%); у Clostridium intestinale (WP_021802294.1, идентичны на 47%); у Clostridiisalibacter paucivorans (WP_026894054.1, идентичны на 36%); и у Bacillus coagulans (WP_061575621.1, идентичны на 42% и находящиеся в COG1104). В контексте данного документа, «функциональный полипептид NifS » представляет собой полипептид NifS который способен функционировать в биосинтезе и/или восстановлении железо-серного (FeS) кластера. Полипептиды NifS были описаны и проанализированы в работах Clausen et al. (2000), Johnson et al. (2005), Olson et al. (2000) и Yuvaniyama et al. (2000).
Полипептид NifU в природных бактериях представляет собой молекулярный каркас полипептида, участвующий в биосинтезе железо-серного (FeS) кластера, для компонентов нитрогеназы. В контексте данного документа, «полипептид NifU» означает полипептид, содержащий аминокислоты, последовательность которых, по меньшей мере, на 31% идентична аминокислотной последовательности, представленной как SEQ ID NO:12, и которая включает домен TIGR02000. Элементы семейства белков домена TIGR02000 специфически участвуют в созревании нитрогеназы. NifU содержит N-концевой домен (pfam01592) и C-концевой домен (pfam01106). Были описаны три разные, но частично гомологичные системы сборки кластеров Fe-S: Isc, Suf и Nif. Система Nif, частью которой является NifU, связана с передачей кластера Fe-S нитрогеназе у ряда азотфиксирующих видов. Гомологи Isc и Suf с эквивалентной доменной архитектурой из Helicobacter и Campylobacter исключены в данном документе из определения NifU. Следовательно NifU специфичен для полипептидов NifU, участвующих в созревании нитрогеназы. Элементы семейства родственных белков домена TIGR01999, которые представляют собой белки IscU (например, из Escherichia coli и Saccharomyces cerevisiae и Homo sapiens), которые содержат гомолог N-концевой области NifU, также исключены в данном документе из определения NifU. Природный полипептид NifU обычно имеет в длину от 260 до 310 аминокислот, а природный мономер имеет молекулярную массу, составляющую примерно 29 кДа. Было идентифицировано большое количество полипептидов NifU, а многочисленные последовательности имеются в общедоступных базах данных. Например, полипептиды NifU, обнаруженные: у Klebsiella michiganensis (Учетный №. WP_049136164.1, идентичны на 97% SEQ ID NO:12); у Klebsiella variicola (WP_050887862.1, идентичны на 90%); у Dickeya solani (WP_057084657.1, идентичны на 80%); у Brenneria goodwinii (WP_048638833.1, идентичны на 73%); у Tolumonas auensis (WP_012728889.1, идентичны на 66%); у Agarivorans gilvus (WP_055731596.1, идентичны на 58%); у Desulfocurvus vexinensis (WP_028587630.1, идентичны на 54%); у Rhodopseudomonas palustris (WP_044417303.1, идентичны на 49%); у Helicobacter pylori (WP_001051984.1, идентичны на 31%) и у Sulfurovum sp. PC08-66 (KIM05011.1, идентичны на 31%). В контексте данного документа, «функциональный полипептид NifU» представляет собой полипептид NifU, который способен функционировать как молекулярный каркас полипептида, участвующий в биосинтезе железо-серного (FeS) кластера. Полипептиды NifU были описаны и проанализированы в работах Hwang et al. (1996), Mühlenhoff et al. (2003) и Ouzounis et al. (1994).
NifS представляет собой пиридоксальфосфат (PLP, витамин B6), зависимую цистеин-десульфуразу, которая генерирует неорганический сульфид, необходимый для синтеза кластера Fe-S из цистеина. В результате реакции образуется аланин, побочный продукт реакции. Реакция протекает через связанный с белком промежуточный продукт цистеин-персульфида, который образуется в результате нуклеофильной атаки высококонсервативного цистеинового остатка (Cys325 в Azotobacter vinelandii) на цистеин-аддукт PLP (Zheng et al., 1994). Сульфид поставляется в NifU для последовательного образования кластеров [Fe2S2] и [Fe4S4]. Фермент NifS действует в бактериях как гомодимер.
NifU обеспечивает каркас для образования кластера [Fe4S4], функционирующего как гомодимер. Полипептид NifU содержит три домена, а именно N-концевой поддерживающий домен, центральный домен и С-концевой поддерживающий домен (Smith et al., 2005). N-концевой домен имеет высокую гомологию последовательностей с белками IscU из бактерий и белками Isu из эукариот, в то время как С-концевой домен гомологичен белкам Nfu, обнаруженным в митохондриях и хлоропластах. Центральный домен содержит один постоянный окислительно-восстановительный кластер [Fe2S2]2+ на субъединицу NifU, который, как полагают, из-за своей стабильности не переносится на другие белки Nif. Считается, что этот кластер координируется четырьмя консервативными цистеиновыми остатками (Cys137, 139, 172 и 175 в NifU A. vinelandii) (Fu et al., 1994). В бактериях NifU образует гомодимер, а его N-концевой домен может связывать один кластер [Fe2S2] на мономер. Кластеры [Fe2S2]в мономерах могут быть слиты с восстановлением и с образованием одного кластера [Fe4S4] на димер NifU. Затем пара кластеров [Fe4S4] доставляется из NifU в NifB и процессируется в ядре 8Fe на NifB, которое впоследствии используется для синтеза FeMo-ко. В дивергентном пути для кластеров Fe-S, один кластер [Fe4S4] , связанный либо с N-концевым, либо с C-концевым каркасным доменом NifU, переносится в апо-NifH для созревания редуктазы нитрогеназы белка NifH (Smith et al., 2005). Было высказано предположение, что NifU также передает два кластера [Fe4S4] белковому комплексу NifD-NifK (обозначенному здесь как стадия 0 D-K), и что NifH конденсирует эту пару кластеров в зрелый P-кластер [Fe8-S7] (Dos Santos et al., 2004). Эти N-концевые кластеры считаются чрезвычайно подвижными и не сохраняются во время очистки (Smith et al., 2005). С-концевой домен может содержать один кластер [Fe4S4] на мономер. В отличие от N-концевого кластера, сборка C концевого кластера [Fe4S4] происходит быстро и не обнаруживается присутствие промежуточного кластера [Fe2S2] (Smith et al., 2005). С-концевые кластеры более стабильны, чем N-концевые кластеры, и их можно сохранять во время очистки. Однако при восстановлении дитионитом, С-концевые кластеры быстро разрушаются (Smith et al., 2005). Используя мутации, заключающиеся в замене цистеина на аланин в NifU, Dos Santos и соавт. показали, что как N-, так и C-концевые кластеры могут быть перенесены в апо-NifH.
В работе López-Torrejón et al. (2016) преведены данные, что белок NifH, способный отдавать электроны голо-NifD-NifK, может генерироваться в митохондриях дрожжей посредством экспрессии как NifH, так и NifM. Эти авторы обнаружили, что в дрожжевых клетках NifS и NifU с этой функцией не требуются, для генерации белка NifH. Они пришли к выводу, что эндогенные пути сборки железо-серного кластера в дрожжевых клетках, предположительно локализованы в митохондриях белков Nfs1 и Nfu1, которые являются родственными белками у дрожжей, и способны передавать кластеры [Fe4S4] в NifH. Следовательно, существует возможность, что NifS и NifU не потребуются для восстановления белка NifH, белка Fe или редуктазы динитрогеназы в дрожжах, но NifS и NifU могут потребоваться для созревания и функционирования NifB и/или NifD-NifK. Неизвестно, обладают ли митохондрии растения аналогичной эндогенной способностью к образованию кластеров [Fe4S4], достаточной для активности нитрогенаы.
Полипептид NifV в при родных бактериях представляет собой гомоцитрат-синтазу (EC 2.3.3.14), продуцирующую гомоцитрат путем переноса ацетильной группы из ацетил-кофермента А (ацетил-СоА) на 2-оксоглутарат. Затем гомоцитрат используется в синтезе FeMo-ко, FeV-ко и FeFe-ко. В контексте данного документа «полипептид NifV» означает полипептид, содержащий аминокислоты, последовательность которых по меньшей мере на 39% идентична аминокислотной последовательности, представленной как SEQ ID NO:13, и которая включает один или оба домена TIGR02660 и DRE_TIM. Элементы семейства белков домена TIGR02660 гомологичны ферментам, которые включают 2-изопропилмалат-синтазу, (R)-цитрамалат-синтазу и гомоцитрат-синтазу, которые связанные с процессами, отличными от азотфиксации. Семейство белков домена cd07939 также включает белки NifV Heliobacterium chlorum и Gluconacetobacter diazotrophicus, которые, по-видимому, ортологичны по отношению к FrbC. Это семейство принадлежит к суперсемейству металлолиаз DRE-TIM. Металлолиазы DRE-TIM включают 2-изопропилмалат-синтазу (IPMS), альфа-изопропилмалат-синтазу (LeuA), 3-гидрокси-3-метилглутарил-СоА-лиазу, гомоцитрат-синтазу, цитрамалат-синтазу, 4-гидрокси-2-оксовалерат-адолазу, рецитрат-синтазу, транскарбоксилазу 5S, пируваткарбоксилазу, AksA и FrbC. Все эти члены имеют долю в консервативном бочкообразном домене триозофосфатизомеразы (TIM), состоящий из основного мотива бета (8) -альфа (8) с восемью параллельными бета-цепями, образующими замкнутый бочонок, окруженный восемью альфа-спиралями. Домен имеет каталитический центр, содержащий сайт связывания двухвалентного катиона, образованный кластером инвариантных остатков, которые покрывают ядро бочонка. Кроме того, каталитический сайт включает три инвариантных остатка - аспартат (D), аргинин (R) и глутамат (E), которые являются основой доменного имени «DRE-TIM». Природный полипептид NifV обычно имеет в длину от 360 до 390 аминокислот, хотя некоторые элементы могут иметь в длину 490 аминокислотных остатков, а природный мономер имеет молекулярную массу, составляющую примерно 41 кДа. Было идентифицировано большое количество полипептидов NifV, а многочисленные последовательности имеются в общедоступных базах данных. Например, полипептиды NifV, обнаруженные: у Klebsiella michiganensis (Учетный №. WP_049083341.1, идентичны на 95% SEQ ID NO:13); у Raoultella ornithinolytica (WP_045858154.1, идентичны на 86%); у Kluyvera intermedia (WP_047370264.1, идентичны на 81%); у Dickeya dadantii (WP_038912041.1, идентичны на 70%); у Brenneria goodwinii (WP_048638835.1, идентичны на 59%); у Magnetococcus marinus (WP_011712856.1, идентичны на 46%); у Sphingomonas wittichii (WP_037528703.1, идентичны на 43%); у Frankia sp. EI5c (OAA29062.1, идентичны на 41%) и у Clostridium sp. Maddingley MBC34-26 (EKQ56006.1, идентичны на 39%). В контексте данного документа, «функциональный полипептид NifV» представляет собой полипептид NifV который способен функционировать как гомоцитрат-синтаза. Полипептиды NifV были описаны и проанализированы в работах Hu et al. (2008), Lee et al. (2000), Masukawa et al. (2007) и Zheng et al. (1997).
Полипептид NifX в Azotobacter vinelandii связывает NifB-ко (Fe6-S9-C), который передается в NifE-NifN для сборки FeMo-ко (Hernandez et al., 2007). Также было показано, что VK-кластеры (Fe8-S9-C или Mo-Fe7-S9-C, Jimenez-Vincente et al., 2015) обмениваются между NifE-NifN, что свидетельствует о его роли как транзиентного резервуара для предшественников FeMo-ко. В работе Hernandez et al. (2007) отмечается, что NifX может действовать как шаперон, который стабилизирует комплексы NifE-NifN или NifD-NifK во время переноса FeMo-ко на апо-NifD-NifK и/или репозиционирует белки в благоприятную ориентацию для переноса FeMo-ко и таким образом действуют, в целях регулирования синтеза FeMo-ко. Активация апо-NifD-NifK экзогенным FeMo-ко комплексами динитрогеназы, экстрагированными из мутантов A. vinelandii , дефицитных по различным комбинациям дополнительных белков NifY/NafY/NifX, показала, что NifX также может помогать FeMo-ко во вставке апо-NifD-NifK. (Rubio et al., 2002). Эта дополнительная функция NifX может быть ответственна за сохранение активности восстановления ацетилена у мутанта Klebsiella ΔnifY, показанная в работе Homer et al. (1993).
Полипептид NifX в природных бактериях представляет собой полипептид, который участвует в синтезе FeMo-ко, по меньшей мере, помогая в переносе предшественников FeMo-ко от NifB к NifE-NifN или FeMo-ко в NifD-NifK. В контексте данного документа, «полипептид NifX» означает полипептид, содержащий аминокислоты, последовательность которых, по меньшей мере, на 29% идентична аминокислотной последовательности, представленной как SEQ ID NO:14, и которая включает один или оба консервативных домена TIGR02663 и cd00853. NifX включен в более крупное семейство белков, связывающих железо-молибденовый кластер, который включает некоторые последовательности NifB и NifY, причем NifX, NafY и С-концевая область некоторых полипептидов NifB содержат домен pfam02579, и каждый из них участвует в синтезе одного или нескольких или всех FeMo-ко, FeV-ко или FeFe-ко. Другие полипептиды NifB, особенно из метаногенных архей и некоторых анаэробных фирмикутов, лишены NifX-подобного домена (Boyd et al., 2011), включая NifB из H. halophila, M. barkeri и C. purinilyticum, упомянутых выше. Некоторые полипептиды NifX были помечены в базах данных как NifY, и наоборот. Природный полипептид NifX, продуцированный сам по себе, а не в результате природного слияния в составе полипептида NifB, обычно имеет в длину от 110 до 160 аминокислот, а природный мономер имеет молекулярную массу, составляющую примерно 15 кДа. Было идентифицировано большое количество полипептидов NifX, а многочисленные последовательности имеются в общедоступных базах данных. Например, полипептиды NifX, обнаруженные: у Klebsiella michiganensis (Учетный №. WP_049070199.1, идентичны на 97% SEQ ID NO:14); у Klebsiella oxytoca (WP_064342937.1, идентичны на 97%); у Raoultella ornithinolytica (WP_044347173.1, идентичны на 91%); у Klebsiella variicola (WP_044612922.1, идентичны на 83%); у Kosakonia radicincitans (WP_043953583.1, идентичны на 75%); у Dickeya chrysanthemi (WP_039999416.1, идентичны на 68%); у Rahnella aquatilis (WP_047608097.1, идентичны на 58%); у, Azotobacter chroococcum (WP_039800848.1, идентичны на 34%); у Beggiatoa leptomitiformis (WP_062149047.1, идентичны на 33%) и у Methyloversatilis discipulorum (WP_020165972.1, идентичны на 29%). В контексте данного документа, «функциональный полипептид NifX» представляет собой полипептид NifX, который способен переносить предшественников FeMo-ко из NifB в NifE-NifN. Полипептиды NifX были описаны и проанализированы в работах Allen et al. (1994) и Shah et al. (1999).
Полипептид NifY в природных бактериях представляет собой полипептид, который участвует в синтезе FeMo-ко, по меньшей мере, помогая в переносе предшественников FeMo-ко от NifB к NifE-NifN. В контексте данного документа, «полипептид NifY» означает полипептид, содержащий аминокислоты, последовательность которых, по меньшей мере, на 34% идентична аминокислотной последовательности, представленной как SEQ ID NO:15, и которая включает один или оба домена TIGR02663 и cd00853. NifY включен в более крупное семейство белков, связывающих железо-молибденовый кластер, который включает NifB и NifX, причем NifX, NafY и С-концевая область NifB все содержат домен pfam02579, и каждый из них участвует в синтезе FeMo-ко. Было идентифицировано большое количество полипептидов NifY, а многочисленные последовательности имеются в общедоступных базах данных. Например, полипептиды NifX, обнаруженные: у Klebsiella michiganensis (Учетный №. WP_049089500.1, идентичны на 99% SEQ ID NO:15); у Klebsiella oxytoca (WP_064342935.1, идентичны на 98%); у Klebsiella quasipneumoniae (WP_044524054.1, идентичны на 90%); у Klebsiella variicola (WP_049010739.1, идентичны на 81% ); у Kluyvera intermedia (WP_047370270.1, идентичны на 69%); у Dickeya chrysanthemi (WP_039999411.1, идентичны на 62%); у Serratia sp. ATCC 39006 (WP_037382461.1, идентичны на 57%); у Rahnella aquatilis (WP_014683024.1, идентичны на 47%); у Pseudomonas putida (AEX25784.1, идентичны на 37%); и у Azotobacter vinelandii (WP_012698835.1, идентичны на 34%). В контексте данного документа, «функциональный полипептид NifY» представляет собой полипептид NifY, который способен переносить предшественников FeMo-ко из NifB в NifE-NifN.
При выделении из мутантных штаммов NifB или NifN-NifE K. oxytoca или A. vinelandii, апо-NifD-NifK связывался с дополнительным полипептидом, называемым γ-белком (Paustian et al, 1990; Homer et al., 1993), с образованием гетерогексамера с полипептидами NifD и NifK (α2β2γ2). Третий полипептид в K. oxytoca кодировался геном NifY (Homer et al., 1993), а добавления очищенного FeMo-ко к очищенному комплексу гетерогексамера α2β2γ2 было достаточно для получения каталитически активной нитрогеназы. Добавление FeMo-ко приводило к диссоциации NifY из комплекса с образованием холофермента (α2β2). Третий полипептид в A. vinelandii кодировался геном NafY (фактор Y, связанный с нитрогеназой; Учетный № AGK13761, Rubio et al., 2002), который отличен, но связан с продуктом гена NifY в A. vinelandii (Учетный № AGK13792). Считалось, что в каждом случае третий полипептид участвует в содействии вставке FeMo-ко с образованием активного фермента. Это подтверждается способностью NafY и NifY связывать FeMo-ко (Homer et al., 1995).
NifY и NafY A. vinelandii связываются с апо-NifD-NifK на разных стадиях созревания холофермента NifD-NifK либо с α-Cys275, либо с α-His442 NifD, оба аминокислотных остатка которых ковалентно служат якорем FeMo-ко (Jimenez- Винсенте и др., 2018). То есть NifY и NafY одновременно не связываются с апо-NifD-NifK. Порядок связывания NifY и NafY с апо-NifD-NifK в настоящее время неизвестен. Диссоциация NifY из NifD-NifK при вставке FeMo-ко была продемонстрирована для нитрогеназы K. oxytoca (Homer et al., 1993) и NafY из NifD-NifK при вставке FeMo-ко для A. vinelandii (Homer et al., 1995). Также полагают, что NafY связывает FeMo-ко через His121 и, возможно, также NifB-ко, что указывает на его роль в качестве FeMo-ко или предшественника инсертазы FeMo-ко (Rubio et al., 2004). NifY A. vinelandii, по-видимому, функционально избыточен из-за отсутствия фенотипа у мутантов ΔnifY (Rubio et al., 2002), и предполагается, что NafY является первичным дополнительным белком к апо-NifD-NifK, который поддерживает вставку FeMo-ко. С другой стороны, виды Klebsiella не имеют гена NafY и имеют только NifY для поддержки вставки FeMo-ко в апо-NifD-NifK, хотя мутант Klebsiella ΔnifY все еще сохранял 60% активности восстановления ацетилена (Homer et al., 1993). Это сохранение функции указывает на присутствие другого вспомогательного белка у Klebsiella, который может частично выполнять функцию NifY в его отсутствие, например, NifX, как описано выше.
В контексте данного документа, «полипептид NafY» означает полипептид, содержащий аминокислоты, последовательность которых, по меньшей мере, идентична на 50% аминокислотной последовательности, представленной как полноразмерная SEQ ID NO:238 (A. vinelandii NafY, Учетный №. AGK13761, 243aa), и которая включает консервативный домен pfam16844. Этот домен, имеющий в длину примерно 91 аминокислотный остаток, обнаруживается сам по себе у некоторых элементов и в аминоконцевой половине более длинных белков NafY. Эта область отрицательно заряжена и, по-видимому, функционирует для распознавания и взаимодействия с апо-NifD-NifK. Природный полипептид NafY обычно имеет в длину от 230 до 250 аминокислот, а природный мономер имеет молекулярную массу, составляющую ~25-28 кДа. Было идентифицировано большое количество полипептидов NafY, а многочисленные последовательности имеются в общедоступных базах данных, причем некоторые из них были аннотированы как полипептиды NifX из-за родства последовательностей NafY и NifX. Например, полипептиды NafY, обнаруженные: у Azotobacter beijerinckii (WP_090728988, идентичны на 93% SEQ ID NO:238); у Pseudomonas stutzeri, (WP_011912501, идентичны на 69%); у Halomonas endophytica (WP_102654474, идентичны на 68%); у Pseudomonas linyingensis (WP_090313081, идентичны на 67%); у Acidihalobacter prosperus (WP_038093031, идентичны на 56%); у Oscillatoriales cyanobacterium (WP_009769409, идентичны на 50%). В контексте данного документа термин «функциональный полипептид NafY» представляет собой полипептид NafY, который способен связываться с апо-NifD-NifK и FeMo-ко. О трехмерной структуре полипептида NafY из A. vinelandii и сравнении и различении последовательностей полипептидов NafY и NifY, NifX, VnfX и NifB сообщалось в работе Dyer et al. (2003).
Полипептид NifZ в природных бактериях представляет собой полипептид, который участвует в синтезе кластера Fe-S, специфически функционируя при связывании второй пары Fe4S4 при образовании второго P-кластера белка MoFe. Считается, что NifZ действует как шаперон, который вызывает конформационные изменения, по меньшей мере, во второй половине белка апо-MoFe, обеспечивая образование второго Р-кластера вместе с NifH. Делеция NifZ в A. vinelandii снижает активность белка MoFe на 66%, но не влияет на активность NifH. В контексте данного документа, «полипептид NifZ» означает полипептид, содержащий аминокислоты, последовательность которых, по меньшей мере, на 28% идентична аминокислотной последовательности, представленной как SEQ ID NO:16, и которая включает консервативный домен pfam04319. Этот домен, имеющий в длину примерно 75 аминокислотный остаток, обнаруживается в изоляции у некоторых элементов и в аминоконцевой половине более длинных белков NifZ. Природный полипептид NifZ имеет в длину от 70 до 150 аминокислот, а природный мономер имеет молекулярную массу, составляющую примерно от 9 до примерно 16 кДа. Было идентифицировано большое количество полипептидов NifZ, а многочисленные последовательности имеются в общедоступных базах данных. Например, полипептиды NifZ обнаруженные: у Klebsiella michiganensis (Учетный №. WP_057173223.1, идентичны на 93% SEQ ID NO:16); у Klebsiella oxytoca (WP_064342939.1, идентичны на 95%); у Klebsiella variicola (WP_043875005.1, идентичны на 77%); у Kosakonia radicincitans (WP_043953588.1, идентичны на 67%); у Kosakonia sacchari (WP_065368553.1, идентичны на 58%); у Ferriphaselus amnicola (WP_062627625.1, идентичны на 47%); у Paraburkholderia xenovorans (WP_011491838.1, идентичны на 41%); у Acidithiobacillus ferrivorans (WP_014029050.1, идентичны на 35%) и у Bradyrhizobium oligotrophicum (WP_015665422.1, идентичны на 28%). В контексте данного документа, «функциональный полипептид NifZ» представляет собой полипептид NifZ который способен связывать кластер Fe4S4 при синтезе кластера Fe-S. Полипептиды NifZ были описаны и проанализированы в работах Cotton (2009) и Hu et al. (2004).
Полипептид NifW в природных бактериях представляет собой полипептид, который связывается с полипептидом NifZ с образованием комплексов более высокого порядка (Lee et al., 1998) и участвует в синтезе или активности белка MoFe (NifD-NifK). NifW и NifZ, по-видимому, участвуют в образовании или накоплении белка MoFe (Paul и Merrick, 1987). В контексте данного документа, «полипептид NifW» означает полипептид, содержащий аминокислоты, последовательность которых, по меньшей мере, идентична на 28% аминокислотной последовательности, представленной как SEQ ID NO:17, и которая содержит консервативный домен белка суперсемейства NifW, идентификационный номер архитектуры 10505077 и принадлежит семейству PF03206. Было идентифицировано большое количество полипептидов NifW, а многочисленные последовательности имеются в общедоступных базах данных. Например, полипептиды NifW, обнаруженные: у Klebsiella oxytoca (Учетный №. WP_064342938.1, идентичны на 98% SEQ ID NO:17); у Klebsiella michiganensis (WP_049080155.1, идентичны на 94%); у Enterobacter sp. 10-1 (WP_095103586.1, идентичны на 90%); у Klebsiella quasipneumoniae (WP_065877373.1, идентичны на 81%); у Pectobacterium polaris (WP_095699971.1, идентичны на 69%); у Dickeya paradisiaca (WP_012764136.1, идентичны на 58%); у Brenneria goodwinii (WP_053085547.1, идентичны на 36%); у Aquaspirillum sp. LM1 (WP_077299824.1, идентичны на 44%); у Candidatus Muproteobacteria bacterium RBG_16_64_10 (OGI40729, идентичны на 34%); у Azotobacter vinelandii (ACO76430.1, идентичны на 32%) и у Methylocaldum marinum (BBA37427.1, идентичны на 28%). Используемый здесь термин «функциональный полипептид NifW» представляет собой полипептид NifW, который способствует или усиливает один или несколько процессов образования, накопления или активности белка MoFe. Функциональный NifW может взаимодействовать с NifZ и/или играть роль в кислородной защите белка MoFe (Gavini et al., 1998).
Большинство организмов, включая бактерии и эукариоты, например растения, имеют многочисленные ферредоксины. Например, в геномах DJ и CA A. vinelandii, соответственно, имеется 15 или 16 белков, аннотированных как ферредоксин или ферредоксин-подобные белки. В контексте данного документа термин «полипептид ферредоксин» представляет собой белок-носитель электронов, имеющий один или два кластера железо-серного типа [2Fe-2S], [3Fe-4S] и/или [4Fe-4S], которые образуют их реакционные центры, см. работы Matsubara и Saeki (1992). Они участвуют во множестве метаболических процессов, включая полипептиды ферредоксина, которые участвуют в фиксации азота, как правило, с более низкой молекулярной массой, чем те, которые не участвуют в нитрогеназе. Основываясь на большом разнообразии ферредоксинов в большинстве клеток и вариациях, наблюдаемых в нескольких исследованиях совместимости или специфичности различных ферредоксинов в дополнение к функции FdxN для синтеза NifB-ко (Yates, 1972; Jimenez-Vincente et al., 2014), наличие ферредоксинов, включая, например, FdxN, лучше всего определять на основе присутствия железо-серных кластеров и их функций, а не на основе аминокислотной идентичности по отношению к стандартной последовательности, например, FdxN A. vinelandii (SEQ ID NO: 232; Учетный № WP_012703542). В контексте данного документа «полипептид FdxN» представляет собой ферредоксин или ферредоксин-подобный полипептид, который функционирует для передачи электронов зрелой редуктазы динитрогеназы NifH и/или для синтеза NifB-ко для нитрогеназы и/или служит промежуточным носителем кластеров [4Fe-4S]. FdxN может функционировать, отдавая электроны зрелой редуктазе динитрогенаы NifH, которая затем передает электроны гетерогексамеру NifD-NifK (см. Yang et al., 2017; Rhizobium japonicum FdxN, Carter et al., 1980; R. meliloti FdxN, Riedel et al. , 1995; Rhodobacter capsulatus FdxN, Jouanneau et al., 1995), или передает электроны полипептиду NifB для синтеза NifB-ко (A. vinelandii: Jimenez-Vincente et al., 2014), или служит промежуточным носителем кластеров [4Fe-4S] (A. vinelandii: Burén et al., 2019) или сочетать любые из этих функций.
Репрезентативными примерами полипептидов FdxN являются примеры, идентифицированные путем поиска в базе данных неизбыточных белков с использованием SEQ ID NO:232 в качестве запроса в BLASTP и показывающие процентную идентичность этой последовательности: Pseudomonas syringae (WP_065835964.1, 85.87%), Candidatus Thiodiazotropha endolucinida (WP_069124666.1, 70,65%), Uliginosibacterium sp. TH139 (WP_101942980, 64,47%), Klebsiella michiganensis (WP_049076934.1, 44,26%), Escherichia coli (WP_072048756.1, 44,26%), Rhizobium leguminosarum (WP_130674512.1, 43,86%) и Flavobacterium alvei (WP_103805005.1, 28,57%).
Идентичность последовательности и замены
Что касается определенного полипептида, то следует понимать, что величины % идентичности, превышающие указанные выше, будут охватывать предпочтительные варианты осуществления. Таким образом, там, где это применимо, в свете величин минимального % идентичности предпочтительно, чтобы полипептид содержал аминокислотную последовательность, которая не менее 30%, более предпочтительно не менее 35%, более предпочтительно не менее 40%, более предпочтительно не менее 45%, более предпочтительно, по меньшей мере, 50%, более предпочтительно, по меньшей мере, 55%, более предпочтительно, по меньшей мере, 60%, более предпочтительно, по меньшей мере, 65%, более предпочтительно, по меньшей мере, 70%, более предпочтительно, по меньшей мере, 75%, более предпочтительно, по меньшей мере, 80%, более предпочтительно, по меньшей мере, 85%, более предпочтительно, по меньшей мере, 90%, более предпочтительно, по меньшей мере, 91%, более предпочтительно, по меньшей мере, 92%, более предпочтительно, по меньшей мере, 93%, более предпочтительно, по меньшей мере, 94%, более предпочтительно, по меньшей мере, 95% , более предпочтительно, по меньшей мере, 96%, более предпочтительно, по меньшей мере, 97%, более предпочтительно, по меньшей мере, 98%, более предпочтительно, по меньшей мере, 99%, более предпочтительно, по меньшей мере, 99,1%, более предпочтительно, по меньшей мере, 99,2%, более предпочтительно, по меньшей мере, 99,3%, более предпочтительно по меньшей мере 99,4%, более предпочтительно по меньшей мере 99,5%, более предпочтительно по меньшей мере 99,6%, более предпочтительно t по меньшей мере 99,7%, более предпочтительно по меньшей мере 99,8% и даже более предпочтительно по меньшей мере 99,9% идентична соответствующей номинированной SEQ ID NO.
Мутантная аминокислотная последовательность полипептидов, определенных в данном документе, может быть получена в результате введения соответствующих нуклеотидных замен в нуклеиновую кислоту, определенную в данном документе, или путем синтеза требуемого полипептида in vitro. Такие мутанты включают, например, одну или несколько делеций, вставок или замен аминокислот. Комбинация мутаций в результате делецией, вставок и замен может использоваться для получения конечной конструкции при условии, что конечный полипептидный продукт обладает требуемыми характеристиками. Предпочтительные мутантные аминокислотные последовательности имеют только одну, две, три, четыре или менее 10 аминокислотных замен по сравнению с эталонным полипептидом дикого типа.
Мутантные (измененные) полипептиды можно получать, используя любую методику, известную в данной области, например, используя стратегии направленной эволюции или рационального конструирования (см. ниже). Продукты, полученные из мутированной/измененной DNA, можно легко подвергнуть скринингу с использованием описанных здесь методик, чтобы определить, изменяет ли их экспрессия в растении его фенотип, по сравнению с соответствующим растением дикого типа, например, приводит ли их экспрессия к увеличению урожайности, биомассы, скорости роста, жизненности, приросту азота, обусловленному биологической фиксацией азота, эффективности использования азота, толерантности к абиотическому стрессу и/или толерантности к недостаточности питательных веществ по сравнению с соответствующим растением дикого типа.
При конструировании мутантной аминокислотной последовательности, локализация сайта мутации и природа мутации будут зависеть от характеристик, которые подлежат изменению. Сайты мутации могут быть изменены индивидуально или последовательно, например, (1) сначала путем замены аминокислот консервативным выбором, а затем более радикальным выбором в зависимости от достигнутых результатов, (2) делецией целевого остатка или (3) вставкой других остатков, прилегающие к сайту локализации.
Делеции аминокислотной последовательности обычно составляют от 1 до 15 остатков, более предпочтительно от 1 до 10 остатков и обычно от 1 до 5 сцепленных остатков.
Мутантное замещение удаляет, по меньшей мере, один аминокислотный остаток в молекуле полипептида и вставляет на его место другой остаток. При необходимости поддержки определенной активности, предпочтительно не производить или производить только консервативные замены в позициях аминокислот, которые являются высококонсервативными в соответствующем семействе белков. Примеры консервативных замен показаны в Таблице 1 под заголовком «примеры замен».
В предпочтительном варианте осуществления мутантный/вариантный полипептид имеет одно, два, три или четыре консервативных аминокислотных замены, по сравнению с природным полипептидом. Подробная информация о консервативных заменах аминокислот представлена в Таблице 1. В предпочтительном варианте осуществления, замены не относятся к одному или нескольким мотивам или доменам, которые являются высококонсервативными среди различных полипептидов, согласно изобретению. Квалифицированному специалисту очевидно, что такие незначительные замены не изменят активность полипептида при экспрессии в рекомбинантной клетке.
Таблица 1. Примеры замен
остаток
замен
Первичная аминокислотная последовательность полипептида, согласно изобретению, может использоваться для конструирования его вариантов/мутантов на основе сравнений с близкородственными полипептидами. Квалифицированному специалисту очевидно, что высококонсервативные остатки среди близкородственных белков могут быть изменены с меньшей вероятностью, особенно с помощью не консервативных замен, при сохранении активности, менее чем у консервативных остатков (см. выше). Более строгим исследованием для идентификации консервативных аминокислотных остатков является выравнивание более отдаленных родственных полипептидов с той же функцией. Для сохранения функции следует поддерживать высококонсервативные остатки, тогда как не консервативные остатки более подвержены заменам или делециям при сохранении функции.
В объем настоящего изобретения также входят полипептиды, которые дифференцированно модифицируются во время или после синтеза в клетке, например, путем гликозилирования, ацетилирования, фосфорилирования или протеолитического расщепления.
Направленное конструирование
Белок может конструироваться направленно на основе известной информации о структуре и укладывании белка. Это достигается путем конструирования с нуля (программируемая конформация белка) или путем вторичного конструирования на основе собственных каркасов (см., например, Hellinga, 1997; и Lu и Berry, Protein Structure Design и Engineering, Handbook of Proteins 2, 1153-1157 (2007)). См., здесь пример 10. Конструирование белков обычно включает в себя идентификацию последовательностей, которые укладываются в заданную или целевую структуру, и может выполняться с использованием компьютерных моделей. Вычислительные алгоритмы конструирования белков ищут в пространстве последовательность-конформация для последовательностей, которые имеют низкую энергию при укладывании в целевую структуру. Вычислительные алгоритмы конструирования белков используют модели белковой энергетики для оценки влияния мутаций на структуру и функцию белка. Эти энергетические функции обычно включают сочетание молекулярной механики, статистических (т. е. основанных на знаниях) и других эмпирических терминов. Подходящим доступным программным обеспечением является программа IPRO (Interative Protein Redesign и Optimization), EGAD (A Genetic Algorithm for Protein Design), Rosetta Design, Sharpen и Abalone.
Линкеры
Используемый здесь в контексте полипептидов термин «линкер» или «олигопептидный линкер» означает одну или несколько аминокислот, которые ковалентно соединяют два или более функциональных домена, например, MTP и NP, два NP, NP и метку. Аминокислоты ковалентно связаны пептидными связями как внутри линкера, так и между линкером и функциональными доменами. Линкер способен обеспечивать свободу перемещения одного функционального домена по отношению к другому, не оказывая существенного вредного воздействия на функцию двух или более доменов. Линкер способствует правильному укладыванию и функционированию одного или обоих функциональных доменов. Специалисту очевидно, что размер линкера может быть определен эмпирически или смоделирован на основе информации об укладывании белка.
Линкер может содержать сайт расщепления протеазой, например МРР. Таким образом, линкер может рассматриваться как часть MTP.
Специалисту очевидно, что C-концевая область MTP может быть трансляционно слита с N-концевой аминокислотой NP без линкера или через линкер из одного или нескольких аминокислотных остатков, например, 1-5 аминокислотных остатков. Таким образом, линкер может рассматриваться как часть MTP.
В некоторых вариантах осуществления, линкер содержит, по меньшей мере, 1 аминокислоту, по меньшей мере, 2 аминокислоты, по меньшей мере, 3 аминокислоты, по меньшей мере, 4 аминокислоты, по меньшей мере, 5 аминокислот, по меньшей мере, 6 аминокислот, по меньшей мере, 7 аминокислот, по меньшей мере, 8 аминокислот, по меньшей мере, 9 аминокислот, по меньшей мере, 10 аминокислот, по меньшей мере, 12 аминокислот, по меньшей мере, 14 аминокислот, минимум 16 аминокислот, минимум 18 аминокислот, по меньшей мере, 20 аминокислот, по меньшей мере, 25 аминокислот, по меньшей мере, 30 аминокислот, по меньшей мере, 35 аминокислот, по меньшей мере, 40 аминокислот, по меньшей мере, 45 аминокислот, по меньшей мере, 50 аминокислот, минимум 60 аминокислот, минимум 70 аминокислот, по меньшей мере, 80 аминокислот, по меньшей мере, 90 аминокислот или примерно 100 аминокислот. В некоторых вариантах осуществления, максимальный размер линкера составляет 100 аминокислот, предпочтительно 60 аминокислот, более предпочтительно 40 аминокислот.
В некоторых вариантах осуществления, линкер позволяет перемещать один функциональный домен по отношению к другому для повышения стабильности слитого полипептида. При необходимости, линкер может включать повторы полиглицина или комбинации остатков глицина, пролина и аланина.
Линкеры для соединения двух полипептидов Nif, например, NifD-линкер-NifK и NifE-линкер-NifN, предпочтительно выбирают по количеству и последовательности аминокислот в линкере, на основании нескольких критериев. Критериями являются: отсутствие цистеинового остатка, чтобы избежать образования нежелательных дисульфидных связей, небольшое количество или предпочтительно отсутствие заряженных остатков (Glu, Asp, Arg, Lys) для снижения вероятности нежелательных взаимодействий поверхностных солевых мостиков, небольшое количество гидрофобных остатков или их отсутствие (Phe, Trp, Tyr, Met, Val, Ile, Leu), поскольку такие остатки могут способствовать проникновению через поверхность полипептида и не имеют аминокислот, которые могут быть посттрансляционно модифицированы. В этом контексте «несколько заряженных остатков» означает менее 10% аминокислотных остатков в линкере, а «несколько гидрофобных остатков» означает менее 15% аминокислотных остатков в линкере.
В одном из вариантов осуществления, линкер не содержит цистеинового остатка.
В одном из вариантов осуществления, линкер содержит четыре, три или два, или один, или не содержит заряженных остатков. Предпочтительно, чтобы в целом линкер содержал четыре, три или два, или один остаток глутаминовой кислоты, аспарагиновой кислоты, аргинина и лизина, либо не содержал их.
В одном из вариантов осуществления, линкер содержит четыре, три или два, или один, или не содержит гидрофобных остатков. Предпочтительно, чтобы в целом линкер содержал четыре, три или два остатка или один остаток фенилаланина, триптофана, тирозина, метионина, валина, изолейцина и лейцина или не содержит их.
В одном из вариантов осуществления, по меньшей мере, 70%, или, по меньшей мере, 80%, или, по меньшей мере, 90% линкера содержат остатки, выбранные из треонина, серина, глицина и аланина.
Обзор использования олигопептидных линкеров для модификации полипептидов представлен в работах Chen et al. (2013) и Zhang et al. (2009).
Метки
В конкретном варианте осуществления слитой полипептид содержит, по меньшей мере, одну метку, подходящую для обнаружения или очистки слитого полипептида или его процессированного продукта. Метка обычно связана с C-концевым или N-концевым доменом слитого полипептида. В предпочтительном варианте осуществления метка связана с С-концевой областью полипептида Nif. Метка обычно представляет собой пептид или аминокислотную последовательность, способную связываться с одной или несколькими лигандами, например, с одной или несколькими лигандами аффинной матрицы, например, хроматографическим носителем или магнитным микроносителем, или антителом с высокой аффинностью. Специалисту очевидно, что метка предпочтительно локализуется в слитом белке в месте, которое не приводит к удалению метки из NP после расщепления MTP после импорта в митохондрии. Кроме того, метка не должна мешать механизму импорта митохондрий. В предпочтительном варианте осуществления, полинуклеотид, согласно изобретению, кодирует слитой полипептид, который содержит, в порядке от N- до C-конца, N-концевой MTP, полипептид Nif и метку обнаружения/очистки. В альтернативном варианте осуществления, слитой полипептид содержит, в порядке от N- до C-конца, N-концевой MTP, метку обнаружения/очистки и полипептид Nif.
Дополнительные иллюстративные, неограничивающие примеры меток, полезных для обнаружения, выделения или очистки слитого полипептида или его процессированного продукта, включают метку гемагглютинина (НА) гриппа человека, гистидиновые метки, содержащие, например, 6 или 8 гистидиновых остатков, флуоресцентные метки, такие как флуоресцеин, резурфин и его производные, Arg-метку, FLAG-метку, Strep-метку, эпитоп, способный распознаваться антителом, такой как c-myc-метка (распознаваемый антителом анти-c-myc), SBP- метка, S-метка, кальмодулин-связывающий пептид, целлюлозно-связывающий домен, хитин-связывающий домен, глутатион-S-трансфераза-метка, мальтозо-связывающий белок, NusA, TrxA, DsbA, Avi-метка и т. д.
Трансляционные слияния с участием полипептидов Nif
В научной литературе описываются осуществленные трансляционные слияния с несколькими полипептидами Nif. Они обобщены в Таблице 2 и в работе Burén и Rubio (2018). Большинство из них включает искусственное добавление эпитопов или связывающих доменов, таких как гистидиновые метки или метки Strep, к белкам обнаружения и очистки, и лишь некоторые из них были экспрессированы в растительных клетках. Имеются сообщения о природном слиянии полипептидов Nif в бактериях. Для анализов на бактериальных хозяевах к NifD добавлялись His-метки различной длины (7-10 гистидинов) (Christiansen et al., 1998), NifE (Goodwin et al., 1998), NifM (Gavini et al., 2006), а также как полноразмерные, так и укороченные версии NifB (Fay et al., 2015). В каждом случае функция Nif сохранялась для модифицированного полипептида Nif, что было продемонстрировано на бактериях или в анализах реконструкции нитрогеназы in vitro.
Таблица 2. Краткое описание слияния генов полипептидов Nif в литературных источниках
Anabaena variabilis
Clostridium pasteruienum
В работе Thiel et al. (1995) идентифицировали природную делецию 29 нуклеотидов и, следовательно, делецию 9 аминокислот и терминирующий кодон NifE в межгенной области между генами NifE и NifN у сине-зеленой водоросли Anabaena variabilis. Делеция привела к слиянию полипептидов NifE-NifN, которое сохранило по меньшей мере некую функцию нитрогеназы полипептидов NifE и NifN. Слитой полипептид NifE-NifN также имел 19 других аминокислотных замен в области слияния, которые могли повлиять на функцию Nif, но неизвестным образом. Ген слияния экспрессировался, но только в строго анаэробных условиях. Не сообщалось, было ли снижение активности по сравнению с не слитыми генами.
В работе Suh et al. (2003) создали искусственное соединение между генами NifD и NifK хромосомы A. vinelandii путем делеции, включающей терминирующий кодон NifD и кодон инициирующий трансляцию (ATG) из NifK, с образованием вектора, обозначенного pBG1404. Делеция привела к чистой потере трех аминокислот и семи аминокислотных замен в аминокислотах 2-10 полипептида NifK. Клетки-хозяева A. vinelandii, содержащие pBG1404, были ослаблены в ходе их роста в среде с низким содержанием азота по сравнению с соответствующими бактериями дикого типа.
В работе Wiig at al. (2011) использовали природное трансляционное слияние генов NifN и NifB, обнаруженное в Clostridium pastuerianum, и определили эту функциональность для активности NifN и NifB в анализах бактериальной и биохимической комплементации. Это слияние было непосредственным, без какого-либо пептидного линкера, т. е. С-концевая область NifN была непосредственно ковалентно связана с N-концевой областью NifB.
В дрожжевых и растительных клетках трансляционные слияния использовались для направления белков, кодируемых в ядре, в митохондриальный матрикс. В тестах на дрожжевую экспрессию было показано, что трансляционные слияния митохондриального целевого пептида (MTP) и некоторых полипептидов Nif (NifH, NifM, NifS и NifU) функционируют при выращивании в аэробных условиях (Lopez-Torrejon et al., 2016). Слияния эпитопов (FLAG и HIS) также оказались функциональными при слиянии с NifH, NifM, NifS и NifU, хотя эти слияния предназначались для локализации в цитоплазме дрожжей и были функциональными только тогда, когда дрожжи росли в анаэробных условиях. В работе Burén et al. (2017b) показано, что версия растворимого варианта NifB, нацеленная на митохондриальный матрикс, была функциональна в анализах комплементации in vitro при повторном выделении из митохондрий дрожжей. Эта версия NifB включала N-концевой MTP, укороченный вариант NifB (без NifX-подобного домена) и С-концевую метку эпитопа 10xHis. Большое количество слияний MTP-Nif также получали в анализе на экспрессию дрожжей. Однако этот большой ряд ко-экспрессированных белков не смог проявить активность в дрожжах (Burén et al., 2017b).
MTP из гена CPN-60 был слит с N-концевой областью NifH, NifM, NifS и NifU и показал свою функциональность в анализах комплементации in vitro, когда Fe-белок был повторно выделен из растений, выращенных при пониженном давлении и при 10% содержании кислорода (US2016/0304842).
Полинуклеотиды
Термины «полинуклеотид» и «нуклеиновая кислота» используются здесь взаимозаменяемо. Они означают полимерную форму нуклеотидов любой длины, дезоксирибонуклеотидов или рибонуклеотидов или их аналогов. Полинуклеотид, определенный здесь, может иметь геномное, cDNA, полусинтетическое или синтетическое происхождение, может быть одноцепочечным или, предпочтительно, двухцепочечным, в силу своего происхождения или манипуляции: (1) не связанным со всем или частью полинуклеотида, с которым он связан в природе (например, полинуклеотид Nif, который не содержит последовательность кодирующую нативный промотор); (2) связанным с полинуклеотидом, отличным от полинуклеотида, который связан в природе (например, полинуклеотид Nif, связанный с нуклеотидной последовательностью, кодирующей MTP, и/или ненативной последовательностью кодирующей промотор); или (3) не встречающийся в природе (например, полинуклеотиды, кодирующие слитые полипептиды MTP-Nif, согласно изобретению). Ниже приведены неограничивающие примеры полинуклеотидов: кодирующие или некодирующие области гена или фрагмента гена, локусы, определенные на основе анализа сцепления, экзоны, интроны, информационная RNA (mRNA), транспортная RNA (tRNA), рибосомальная RNA (rRNA), рибозимы, cDNA, рекомбинантные полинуклеотиды, разветвленные полинуклеотиды, плазмиды, векторы, выделенная DNA любой последовательности, выделенная RNA любой последовательности, химерная DNA любой последовательности, зонды на основе нуклеиновой кислоты и праймеры. Полинуклеотид может содержать модифицированные нуклеотиды, например, метилированные нуклеотиды и аналоги нуклеотидов. При наличии, модификации нуклеотидной структуры могут быть внесены до или после сборки полимера. Последовательность нуклеотидов может быть прервана ненуклеотидными компонентами. Полинуклеотид можно дополнительно модифицировать после полимеризации, например, путем конъюгации с метящим компонентом.
«Выделенный полинуклеотид» по существу не содержит компонентов, которые обычно связаны (напр., регуляторные последовательности) или объединяются с полинуклеотидом. Таким образом, выделенный полинуклеотид практически не содержит другого клеточного материала или культуральной среды при продуцировании рекомбинантными методами или практически не содержит химических предшественников или других химикатов при химическом синтезе. Предпочтительно, чтобы выделенный полинуклеотид был свободен, по меньшей мере, на 60%, более предпочтительно, по меньшей мере, на 75% и более предпочтительно, по меньшей мере, на 90% от указанных компонентов.
В контексте данного документа, фраза «экзогенный полинуклеотид» относится к полинуклеотиду, последовательность которого начинается вне клетки или организма, в которых присутствует экзогенный полинуклеотид.
В контексте данного документа, термин «ген» следует понимать в самом широком контексте, он охватывает дезоксирибонуклеотидные последовательности, содержащие транскрибируемую область, а в случае трансляции, кодирующую белок область структурного гена, включая последовательности, локализованные рядом с кодирующей областью на обоих концах 5’ и 3’, и находящиеся на расстоянии, составляющем, по меньшей мере, примерно в 2 т.п.н. на каждом конце и которые участвуют в экспрессии гена. В этом отношении ген включает в себя сигналы управления, например, промоторы, энхансеры, сигналы терминации трансляции и транскрипции и/или сигналы полиаденилирования, которые естественным образом связаны с данным геном, или гетерологичные сигналы управления, и в этом случае ген называется «химерный ген». Последовательности, которые локализованы на 5’ кодирующей белок области и присутствуют в mRNA, называются 5’-нетранслируемыми последовательностями. Последовательности, которые локализованы на 3’ кодирующей белок области или ниже и присутствуют в mRNA, называются 3’-нетранслируемыми последовательностями. Термин «ген» охватывает как cDNA, так и геномные формы гена. Геномная форма или клон гена содержит кодирующую область, которая может быть прервана некодирующими последовательностями, называемыми «интронами», «промежуточными областями» или «промежуточными последовательностями». Интроны представляют собой сегменты гена, которые транскрибируются в ядерную RNA (nRNA). Интроны могут содержать регуляторные элементы, например, энхансеры. Интроны удаляются или «сплайсируются» из ядерного или первичного транскрипта, поэтому интроны отсутствуют в транскрипте mRNA. Функция mRNA во время трансляции определяет последовательность или порядок аминокислот в формирующемся полипептиде. Термин «ген» охватывает синтетическую или слитую молекулу, кодирующую все или часть белков, согласно изобретению, описанных в данном документе, и нуклеотидную последовательность, комплементарную любому из вышеперечисленного.
В контексте данного документа, «химерная DNA», также называемая здесь «конструкцией DNA», означает любую молекулу DNA, которая не встречается в природе, но которая искусственно соединяет две части DNA в единую молекулу, каждая часть которой может быть обнаружена в природе, но целой в природе нет. Например, конструкция DNA, кодирующая слитой полипептид MTP-Nif согласно изобретению. Обычно химерная DNA содержит регуляторные и транскрибируемые или кодирующие белок последовательности, которые вместе в природе не встречаются (напр., полинуклеотид Nif, связанный с ненативной последовательностью, кодирующей промотор). Соответственно, химерная DNA может содержать регуляторные последовательности и кодирующие последовательности, происходящие из разных источников, или регуляторные последовательности и кодирующие последовательности, происходящие из одного и того же источника, но расположенные иначе, чем в природе. Открытая рамка считывания может быть связана, а может и не быть связана с ее природными расположенными выше и ниже регуляторными элементами. Открытая рамка считывания может быть встроена, например, в геном растения в не природной локации или в репликон, или вектор, где она не встречается в природных условиях, например в бактериальной плазмиде или вирусном векторе. Термин «химерная DNA» не ограничивается молекулами DNA, которые реплицируются в хозяине, но включает DNA, способную лигироваться в репликон, например, с помощью специфичных адаптерных последовательностей.
«Трансген» представляет собой ген, который был введен в геном с помощью процедуры трансформации. Термин охватывает ген в клетке-потомке, растении, семени, нечеловеческом организме или его части, который был введен в геном их клетки-предшественника. Такие клетки-потомки и т. д. могут быть потомством, по меньшей мере, 3-го или 4-го поколения клетки-предшественника, которая была первично трансформированной клеткой. Потомство может быть получено половым или вегетативным способом, например, из клубней картофеля или отростков от корня сахарного тростника. Термин «генетически модифицированный» и его варианты являются более широким термином, который охватывает введение гена в клетку путем трансформации или трансдукции, мутацию гена в клетке и генетическое изменение или модуляцию регуляции гена в клетке, или потомство любой клетки, модифицированной, описанной выше способом.
В контексте данного документа, термин «геномная область» относится к позиции в геноме, куда трансген или группа трансгенов (также называемая здесь кластером) были вставлены в клетку или ее предшественника. Такие области содержат только нуклеотиды, которые были встроены вмешательством человека, например, способами, описанными в данном документе.
«Рекомбинантный полинуклеотид», согласно изобретению, относится к молекуле нуклеиновой кислоты, которая была сконструирована или модифицирована с помощью способов искусственной рекомбинации. Рекомбинантный полинуклеотид может присутствовать в клетке в измененном количестве или экспрессироваться с измененной скоростью (напр., в случае mRNA) по сравнению с его нативным состоянием. В одном варианте осуществления, полинуклеотид вводят в клетку, которая в природе не содержит полинуклеотид. Обычно экзогенная DNA используется в качестве матрицы для транскрипции mRNA, которая затем транслируется в непрерывную последовательность аминокислотных остатков, кодирующих полипептид, согласно изобретению, в трансформированной клетке. В другом варианте осуществления, полинуклеотид является эндогенным для бактериальной клетки, а его экспрессия изменяется с помощью средств рекомбинации, например, экзогенная регуляторная последовательность вводится выше эндогенного исследуемого гена, чтобы трансформированная клетка могла экспрессировать полипептид, кодируемый геном.
Рекомбинантный полинуклеотид, согласно изобретению, включает полинуклеотиды, которые не были отделены от других компонентов клеточной или безклеточной системы экспрессии, в которой он присутствует, и включает полинуклеотиды, продуцированные в указанных клеточных или безклеточных системах, которые впоследствии очищены, по меньшей мере, от некоторых других компонентов. Полинуклеотид может представлять собой сцепленной участок нуклеотидов, существующих в природе (напр., полинуклеотид Nif), или включать два или несколько сцепленных участка нуклеотидов из разных источников (встречающихся в природе и/или синтетических), соединенных с образованием одного полинуклеотида (напр., полинуклеотид Nif, связанный с нуклеотидной последовательностью, кодирующей MTP, и/или ненативной последовательностью кодирующей промотор). Обычно такие химерные полинуклеотиды содержат, по меньшей мере, открытую рамку считывания, кодирующую полипептид, согласно изобретению, функционально связанный с промотором, подходящим для управления транскрипцией открытой рамки считывания в исследуемой клетке. Ссылка на «промотор» в данном документе охватывает один промотор или несколько промоторов.
Что касается определенного полинуклеотида, то следует понимать, что величины % идентичности, превышающие указанные выше, будут охватывать предпочтительные варианты осуществления. Таким образом, там, где это применимо, в свете величин минимального % идентичности предпочтительно, чтобы полинуклеотид содержал полинуклеотидную последовательность, которая, по меньшей мере, 60%, более предпочтительно, по меньшей мере, 65%, более предпочтительно, по меньшей мере, 70%, более предпочтительно, по меньшей мере, 75%, более предпочтительно, по меньшей мере, 80%, более предпочтительно, по меньшей мере, 85%, более предпочтительно, по меньшей мере, 90%, более предпочтительно, по меньшей мере, 91%, более предпочтительно, по меньшей мере, 92%, более предпочтительно, по меньшей мере, 93%, более предпочтительно, по меньшей мере, 94%, более предпочтительно, по меньшей мере, 95% , более предпочтительно, по меньшей мере, 96%, более предпочтительно, по меньшей мере, 97%, более предпочтительно, по меньшей мере, 98%, более предпочтительно, по меньшей мере, 99%, более предпочтительно, по меньшей мере, 99,1%, более предпочтительно, по меньшей мере, 99,2%, более предпочтительно, по меньшей мере, 99,3%, более предпочтительно по меньшей мере 99,4%, более предпочтительно по меньшей мере 99,5%, более предпочтительно по меньшей мере 99,6%, более предпочтительно t по меньшей мере 99,7%, более предпочтительно по меньшей мере 99,8% и даже более предпочтительно по меньшей мере 99,9% идентична соответствующей номинированной SEQ ID NO.
Полинуклеотид, согласно изобретению, или полезный для него, может выборочно создавать, при жестких требованиях, гибриды с полинуклеотидом, определенным в данном документе. В контексте данного документа, жесткие требования представляют собой: (1) использование во время гибридизации денатурирующего агента, например, формамида, например, 50% (об/об) формамида с 0,1% (мас./об) бычьего сывороточного альбумина, 0,1% фиколла, 0,1% поливинилпирролидона, 50 ммоль натрий-фосфатного буфера при pH 6,5 с 750 ммоль NaCl, 75 ммоль цитрата натрия при 42 °C; или (2) использование 50%формамида, 5 x SSC (0,75 моль/л NaCl, 0,075 моль/л цитрата натрия), 50 ммоль фосфата натрия (pH 6,8), 0,1% пирофосфата натрия, раствор Денхардта 5x, обработанную ультразвуком DNA сперму лосося (50 г/мл), 0,1% SDS и 10% сульфата декстрана при 42°C в 0,2 x SSC и 0,1% SDS, и/или (3) использование раствора низкой ионной силы и высокой температуры для промывки, например, 0,015 моль/л NaCl/0,0015 моль/л цитрата натрия/0,1% SDS при 50°C.
Полинуклеотиды, согласно изобретению, могут обладать, по сравнению с природными молекулами, одной или несколькими мутациями, которые представляют собой делеции, вставки или замены нуклеотидных остатков. Полинуклеотиды, которые имеют мутации, по сравнению с эталонной последовательностью, могут быть природными (то есть, выделенными из природного источника), либо синтетическими (полученными, например, путем проведения сайт-направленного мутагенеза или перестановки DNA в нуклеиновой кислоте, как описано выше).
Полинуклеотиды, согласно изобретению, могут быть кодон-модифицированы для экспрессии в растительной клетке. Специалисту очевидно, что кодирующая белок область может быть кодон-оптимизирована относительно, например, кодирующей области встречающегося в природе полинуклеотида в азотфиксирующей бактерии.
Конструкции нуклеиновых кислот
Настоящее изобретение относится к конструкциям нуклеиновых кислот, содержащих один или несколько полинуклеотидов, согласно изобретению, а также к векторам и клеткам-хозяевам их содержащим, и к способам их продуцирования и применения. Настоящее изобретение относится к элементам, которые функционально соединены или связаны. Термины «функционально соединенный» или «функционально связанный» и т. п. относятся к функциональной взаимосвязи полинуклеотидных элементов. Обычно функционально соединенные последовательности нуклеиновой кислоты сцепляются где необходимо для соединения двух, сцепленных в рамке считывания, кодирующих белок областей. Кодирующая последовательность «функционально соединится» с другой кодирующей последовательностью, если RNA-полимераза будет транскрибировать две кодирующие последовательности в единую RNA, которая при трансляции затем транслируется в один полипептид, имеющий аминокислоты, полученные из обеих кодирующих последовательностей. Кодирующие последовательности не обязательно должны быть сцеплены друг с другом, пока экспрессируемые последовательности, в конечном итоге, процессируются для получения требуемого белка.
В контексте данного документа, термин «цис-действующая последовательность», «цис-действующий элемент» или «цис-регуляторная область», или «регуляторная область» или аналогичный термин означает любые последовательности нуклеотидов, которые при правильном позиционировании и соединении относительно экспрессируемой генетической последовательности, способны регулировать, по меньшей мере, частично, экспрессию генетической последовательности. Специалистам в данной области очевидно, что цис-регуляторная область способна активировать, подавлять, усиливать, репрессировать или иным образом изменять уровень экспрессии и/или специфичность к клеточному типу, и/или специфичность к развитию последовательности гена на транскрипционном или посттранскрипционном уровне. В предпочтительных вариантах осуществления настоящего изобретения, цис-действующая последовательность представляет собой активирующую последовательность, которая усиливает или стимулирует экспрессию экспрессируемой генетической последовательности.
Термин «оперативное соединение» промотора или энхансерного элемента с транскрибируемым полинуклеотидом означает помещение транскрибируемого полинуклеотида (напр., кодирующего белок полинуклеотида или другого транскрипта) под контроль регуляции промотора, который затем ругулирует транскрипцию этого полинуклеотида. При конструировании комбинаций гетерологичного промотора/структурного гена обычно является предпочтительным позиционирование промотора или его варианта на расстоянии от сайта начала транскрипции транскрибируемого полинуклеотида, которое приблизительно равно расстоянию между этим промотором и кодирующей белок областью, которую он контролирует в своей естественной обстановке, то есть гена, из которого получен промотор. Как известно в данной области техники, некоторое изменение этого расстояния может выполняться без потери функции. Точно так же, предпочтительное позиционирование элемента регуляторной последовательности (напр., оператора, энхансера и т.д.) по отношению к транскрибируемому полинуклеотиду, который должен находиться под его регуляцией, определяется позиционированием элемента в его естественных условиях, т. е. гена, из которого он получен.
В контексте данного документа, термин «промотор» или «промоторная последовательность» относится к области гена, обычно расположенной выше (5’) области, кодирующей RNA, которая контролирует инициирование и уровень транскрипции в исследуемой клетке. Термин «промотор» охватывает регуляторные последовательности транскрипции классического геномного гена, такие как последовательности TATA-бокса и CCAAT-бокса, а также дополнительные регуляторные элементы (напр., активирующие последовательности, расположенные выше, энхансеры и сайленсеры), которые изменяют экспрессию гена в ответ на развитие и/или стимулы окружающей среды, или тканеспецифичным или специфичным для клеточного типа образом. Промотор обычно, но не обязательно, (например, некоторые промоторы PolIII), расположен выше структурного гена, экспрессию которого он регулирует. Кроме того, регуляторные элементы, содержащие промотор, обычно позиционируются в пределах 2 т.п.н. от сайта начала транскрипции гена. Промоторы могут содержать дополнительные специфичные регуляторные элементы, локализованные дальше от сайта начала, для дальнейшего усиления экспрессии в клетке и/или для изменения времени или индуцибельности экспрессии структурного гена, с которым он функционально связан.
Термин «конститутивный промотор» относится к промотору, который управляет экспрессией функционально связанной транскрибируемой последовательности во многих или во всех тканях организма, например, растения. В контексте данного документа, термин «конститутивный» не обязательно означает, что ген экспрессируется на одном уровне во всех типах клеток, но означает, что ген экспрессируется в широком диапазоне типов клеток, хотя часто обнаруживаются некоторые вариации в уровне экспрессии. В контексте данного документа, термин «селективная экспрессия» относится к экспрессии почти исключительно в специфичных органах, например, растении, например, эндосперме, зародыше, листьях, плодах, клубнях или корне. В предпочтительном варианте осуществления, промотор избирательно или предпочтительно экспрессируется в корнях, листьях и/или стеблях растения, предпочтительно злакового растения. Таким образом, избирательная экспрессия может быть противопоставлена конститутивной экспрессии, которая относится к экспрессии во многих или во всех тканях растения в большинстве или во всех условиях среды, в которых находится растение.
Селективная экспрессия может также приводить к компартментации продуктов экспрессии генов в специфичных тканях, органах растений или стадиях развития. Компартментация в определенных субклеточных локациях, таких как пластида, цитозоль, вакуоль или апопластное пространство, может достигаться в результате включения в структуру генного продукта соответствующих сигналов, напр., сигнального пептида для транспорта в требуемый клеточный компартмент или, в случае полуавтономных органелл (пластиды и митохондрии), путем интеграции трансгена с соответствующими регуляторными последовательностями непосредственно в геном органеллы.
Термин «тканеспецифичный промотор» или «органспецифичный промотор» относится к промотору, который предпочтительно экспрессируется в одной ткани или органе, по сравнению со многими другими тканями или органами, предпочтительно в большинстве, если не во всех других тканях или органах, например, в растении. Обычно промотор экспрессируется на уровне в 10 раз выше в специфичной ткани или органе, чем в других тканях или органах.
В одном из вариантов осуществления, промотор представляет собой специфичный для стебля промотор, специфичный для листа промотор или промотор, который управляет экспрессией гена в надземной части растения (по меньшей мере, в стеблях и листьях) (промотор, специфичный для ткани зеленых листьев), например, рибулозо-1,5-бифосфаткарбоксилаза/оксигеназы (RUBISCO).
Примеры промоторов, специфичных для стебля, включают, не ограничиваясь, промоторы, описанные в патенте US 5625136.
В одном варианте осуществления, промотор представляет собой промотор, специфичный для корня. Примеры промоторов, специфичные для корня, включают, не ограничиваясь, промотор для гена кислой хитиназы и специфичные субдомены промотора 35S CaMV.
Промоторы, предусмотренные настоящим изобретением, могут быть нативными для трансформируемого растения-хозяина или могут происходить из альтернативного источника, где область является функциональной в растении-хозяине. Другие источники включают гены T-DNA Agrobacterium, например, промоторы генов для биосинтеза нопалина, октапина, маннопина или других промоторов опина, тканеспецифичные промоторы (см., напр., патент US 5 459 252 и WO 91/13992); промоторы из вирусов (включая вирусы, специфичные для хозяина), и частично или полностью синтетические промоторы. Многочисленные промоторы, которые функциональны в односемядольных и двусемядольных растениях, хорошо известны в данной области (см., напр., Salomon et al., 1984; Garfinkel et al., 1983; Barker et al., 1983), включая различные промоторы, выделенные из растений и вирусов, например, промотор вируса мозаики цветной капусты (CaMV 35S, 19S). Неограничивающие способы оценки активности промотора раскрыты в работах Medberry et al. (1992, 1993), Sambrook et al. (1989, выше) и US 5 164 316.
Альтернативно или дополнительно промотор может быть индуцибельным промотором или промотором, регулируемым в процессе развития, который способен управлять экспрессией введенного полинуклеотида на соответствующей стадии развития, например, растения. Другие цис-действующие последовательности, которые можно использовать, включают энхансеры транскрипции и/или трансляции. Энхансерные области хорошо известны специалистам в данной области и могут включать кодон инициации трансляции ATG и соседние последовательности. При включении кодона инициации, он должен находиться в фазе с рамкой считывания кодирующей последовательности, относящейся к чужеродному или экзогенному полинуклеотиду, чтобы гарантировать трансляцию всей последовательности, если она подлежит трансляции. Области инициации трансляции могут быть получены из источника области инициации транскрипции или из чужеродного или экзогенного полинуклеотида. Последовательность также может происходить из источника промотора, выбранного для управления транскрипцией, а может быть специально модифицирована для увеличения трансляции mRNA.
Конструкция нуклеиновой кислоты, согласно настоящего изобретения, может содержать 3’-нетранслируемую последовательность из примерно 50-1000 нуклеотидных пар оснований, которая может включать последовательность терминации транскрипции. 3’-нетранслируемая последовательность может содержать сигнал терминации транскрипции, который может включать или не включать сигнал полиаденилирования и любые другие регуляторные сигналы, способные влиять на процессинг mRNA. Сигнал полиаденилирования действует для добавления трактов полиадениловой кислоты к 3’-концу предшественника mRNA. Сигналы полиаденилирования обычно распознаются по наличию гомологии с канонической формой 5’ AATAAA-3’, хотя нередки и вариации. Последовательности терминации транскрипции, которые не включают сигнал полиаденилирования, включают терминаторы для PolI или PolIII RNA-полимеразы, которые содержат серию из четырех или более тимидинов. Примерами подходящих 3’-нетранслируемых последовательностей являются 3’-транскрибируемые нетранслируемые области, содержащие сигнал полиаденилирования от гена октопинсинтазы (ocs) или гена нопалинсинтазы (nos) Agrobacterium tumefaciens (Bevan et al., 1983). Подходящие 3’-нетранслируемые последовательности также могут происходить из генов растений, например, гена рибулозо-1,5-бифосфаткарбоксилаза/оксигеназы (ssRUBISCO), хотя также могут использоваться другие 3’-элементы, известные специалистам в данной области.
Поскольку последовательность DNA, вставленная между сайтом инициации транскрипции и началом кодирующей последовательности, т. е. нетранслируемая 5’-лидерная последовательность (5’UTR), может влиять на экспрессию гена, если она транслируется, а также транскрибируется, то может использоваться конкретная лидерная последовательность. Подходящие лидерные последовательности включают те последовательности, которые содержат последовательности, выбранные для управления оптимальной экспрессией чужеродной или эндогенной последовательности DNA. Например, такие лидерные последовательности включают, предпочтительную, обобщающую типичную последовательность, которая может увеличивать или сохранять стабильность mRNA и предотвращать несоответствующую инициацию трансляции, как, например, описано работе Joshi (1987).
Векторы
Настоящее изобретение относится к использованию векторов для манипуляции или переноса генетических конструкций. Вектор представляет собой молекулу нуклеиновой кислоты, предпочтительно молекулу DNA, которую можно использовать для искусственного переноса чужеродного генетического материала в другую клетку, где он может реплицироваться или экспрессироваться. Вектор, содержащий чужеродную DNA, именуется «рекомбинантным вектором». Примеры векторов включают, не ограничиваясь, плазмиды, вирусные векторы, космиды, внехромосомные элементы, минихромосомы, искусственные хромосомы. Вектор может содержать подвижный элемент.
Вектор предпочтительно представляет собой двухцепочечную DNA и содержит один или несколько уникальных сайтов рестрикции, и может быть способен к автономной репликации в определенной клетке-хозяине, включая клетку-мишень или ткань, или их клетку-предшественницу, или ее ткань, или может быть способен интегрироваться в геном, предпочтительно, ядерный геном определенного хозяина, делая клонированную последовательность воспроизводимой. Соответственно, вектор может быть автономно реплицирующимся вектором, т. е. вектором, который существует как внехромосомный объект, репликация которого не зависит от хромосомной репликации, например линейной или замкнутой кольцевой плазмидой, внехромосомным элементом, минихромосомой или искусственной хромосомой. Вектор может содержать любые средства для обеспечения саморепликации. Альтернативно, вектор может быть вектором, который при введении в клетку интегрируется в геном, предпочтительно ядерный геном клетки-реципиента и реплицируется вместе с хромосомой (ами), в которую он был интегрирован. Векторная система может включать один вектор или плазмиду, два или более векторов или плазмид, которые вместе содержат всю DNA, которая подлежит введению в клетку-хозяина или транспозон. Выбор вектора обычно будет зависеть от совместимости вектора с клеткой, в которую должен вводиться вектор. Вектор может также включать маркер отбора, например, ген устойчивости к антибиотикам, ген устойчивости к гербицидам или другой ген, который можно использовать для отбора подходящих трансформантов. Примеры таких генов хорошо известны специалистам в отрасли техники, к которой относится данное изобретение.
Конструкцию нуклеиновой кислоты, согласно изобретению, можно ввести в вектор, такой как плазмида. Векторные плазмиды обычно включают дополнительные последовательности нуклеиновых кислот, которые обеспечивают простой выбор, амплификацию и трансформацию кассеты экспрессии в прокариотических и эукариотических клетках, например, векторы pUC-производные, векторы pSK-производные, векторы pGEM-производные, векторы pSP-производные, векторы pBS-производные, или бинарные векторы, содержащие одну или несколько областей T-DNA. Дополнительные последовательности нуклеиновых кислот включают: точки начала репликации для обеспечения автономной репликации вектора; селективные маркерные гены, предпочтительно кодирующие устойчивость к антибиотикам или гербицидам; уникальные множественные сайты клонирования, обеспечивающие множественные сайты для вставки последовательностей нуклеиновых кислот или генов, кодируемых в конструкции нуклеиновой кислоты; и последовательности, которые усиливают трансформацию прокариотических и эукариотических растительных клеток (особенно).
Под «маркерным геном» подразумевается ген, который придает особый фенотип клеткам, экспрессирующим маркерный ген, и, таким образом, позволяет отличать такие трансформированные клетки от клеток, не имеющих маркера. Ген селектируемого маркера придает признак, по которому можно осуществлять «отбор» на основании устойчивости к селективному агенту (напр., гербициду, антибиотику, радиации, нагреванию или другой обработке, повреждающей нетрансформированные клетки). Маркерный ген, подлежащий скринингу, (или репортерный ген) придает признак, который можно идентифицировать посредством наблюдения или исследования, т. е. посредством «скрининга» (напр., β-глюкуронидазы, люциферазы, GFP или активность другого фермента, отсутствующего в нетрансформированных клетках). Маркерный ген и исследуемая нуклеотидная последовательность не обязательно должны быть связаны.
Для облегчения идентификации трансформантов, конструкция нуклеиновой кислоты должная содержать селектируемый или скринабельный маркерный ген в качестве чужеродного или экзогенного полинуклеотида или в дополнение к нему. Фактический выбор маркера не имеет решающего значения, если он является функциональным (т.е. селективным) в сочетании с клеткой-хозяином, предпочтительно с клеткой-хозяином растения. Маркерный ген и исследуемый чужеродный или экзогенный полинуклеотид не обязательно должны быть связаны, поскольку совместная трансформация несвязанных генов, как, например, описанная в патенте США 4 399 216, также является эффективным процессом трансформации растений.
Примерами селектируемых бактериальных маркеров являются маркеры, которые придают устойчивость к антибиотикам, например, устойчивость к ампициллину, эритромицину, хлорамфениколу или тетрациклину и, предпочтительно, устойчивость к канамицину. Примеры селектируемых маркеров для отбора трансформантов растений включают, не ограничиваясь: ген hyg, который кодирует устойчивость к гигромицину B; ген (nptII) неомицинфосфотрансферазы, придающий устойчивость к канамицину, паромомицину, G418; ген глутатион-S-трансферазы из печени крысы, придающий устойчивость к гербицидам, полученным из глутатиона, как, например, описано в патенте EP 256223; ген глутамин синтетазы, придающий при сверхэкспрессии устойчивость к ингибиторам глутамин синтетазы, таким как фосфинотрицин, как, например, описанное в WO 87/05327; ген ацетилтрансферазы из Streptomyces viridochromogenes, придающий устойчивость к селективному агенту фосфинотрицину, как, например, описано в EP 275957; ген, кодирующий 5-енолшикимат-3-фосфатсинтазу (EPSPS), придающий устойчивость к N-фосфонометилглицину, как, например, описано в работе Hinchee et al. (1988); ген bar, придающий устойчивость к биалафосу, как, например, описано в WO91/02071; ген нитрилазы, такой как bxn из Klebsiella ozaenae, который придает устойчивость к бромоксинилу (Stalker et al., 1988); ген дигидрофолатредуктазы (DHFR), придающий устойчивость к метотрексату (Thillet et al., 1988); мутантный ген ацетолактатсинтазы (ALS), который придает устойчивость к имидазолинону, сульфонилмочевине или другим химическим веществам, ингибирующим ALS (EP 154204); мутировавший ген антранилатсинтазы, придающий устойчивость к 5-метилтриптофану; или ген далапондегалогеназы, придающий устойчивость к гербициду.
Предпочтительные скринабельные маркеры включают, не ограничиваясь: ген uidA, кодирующий фермент β-глюкуронидазы (GUS), для которого известны различные хромогенные субстраты; ген β-галактозидазы, кодирующий фермент, для которого известны хромогенные субстраты; ген акворина (Prasher et al., 1985), который можно использовать для определения биолюминесценции, чувствительной к кальцию; ген зеленого флуоресцентного белка (Niedz et al., 1995) или его производные; ген люциферазы (luc) (Ow et al., 1986), который позволяет детектировать биолюминесценцию, и другие гены, известные в данной области. В настоящем раскрытии, под «репортерной молекулой» подразумевается молекула, которая по своей химической природе обеспечивает аналитически идентифицируемый сигнал, который облегчает определение активности промотора по отношению к белковому продукту.
Предпочтительно, конструкция нуклеиновой кислоты стабильно встроена в геном, например, растения. Соответственно, нуклеиновая кислота содержит соответствующие элементы, которые позволяют молекуле быть включенной в геном, или конструкция помещается в соответствующий вектор, который может быть встроен в хромосому растительной клетки.
В одном варианте осуществления настоящего изобретения, используется рекомбинантный вектор, который содержит, по меньшей мере, один полинуклеотид, определенный в данном документе, и способный доставлять полинуклеотид в клетку-хозяина. Такой вектор содержит гетерологичные последовательности нуклеиновых кислот, то есть последовательности нуклеиновых кислот, которые в природе не обнаруживаются рядом с молекулами нуклеиновых кислот по настоящему изобретению и которые предпочтительно происходят от видов, отличных от видов, из которых происходит молекула (ы) нуклеиновой кислоты. Вектор может представлять собой либо RNA или DNA, либо прокариотический вектор или эукариотический вектор, и обычно представляет собой вирус или плазмиду.
Рекомбинантные векторы, согласно изобретению, содержат слитые последовательности, которые приводят к экспрессии молекул нуклеиновых кислот в виде слитых белков.
Рекомбинантные векторы могут также включать промежуточные и/или нетранслируемые последовательности, окружающие и/или находящиеся внутри последовательности нуклеиновой кислоты полинуклеотида, определенного в данном документе.
Предпочтительно, рекомбинантный вектор стабильно встроен в геном клетки-хозяина, например, растительной клетки. Соответственно, рекомбинантный вектор может содержать соответствующие элементы, которые позволяют встраивать вектор в геном или в хромосому клетки.
Рекомбинантные клетки
Другой вариант осуществления настоящего изобретения относится к рекомбинантной клетке, например, рекомбинантной растительной клетке, которая является клеткой-хозяином, трансформированной одним или несколькими полинуклеотидами, конструкциями или векторами по настоящему изобретению, или их клетками-потомками. В контексте данного документа, термин «рекомбинантная клетка» используется здесь взаимозаменяемо с термином «трансгенная клетка».
Трансформация молекулы нуклеиновой кислоты в клетку может осуществляться любым способом, в результате которого молекула нуклеиновой кислоты может встраиваться в клетку. Методы трансформации включают, не ограничиваясь, трансфекцию, электропорацию, микроинъекцию, липофекцию, адсорбцию и слияние протопластов. Рекомбинантная клетка может оставаться одноклеточной или может вырасти в ткань, орган или многоклеточный организм. Трансформированные молекулы нуклеиновой кислоты, согласно изобретению, могут оставаться внехромосомными или могут интегрироваться в один или несколько участков хромосомы трансформированной клетки таким образом, что сохраняется их способность к экспрессии.
Предпочтительными клетками-хозяевами являются клетки растений, более предпочтительно клетки злаковых растений, более предпочтительно клетки ячменя или пшеницы и даже более предпочтительно клетки пшеницы.
Рекомбинантная клетка может быть культуральной клеткой, клеткой in vitro или клеткой в организме, например, растении, или в органе, например, корне, листе или стебле. Предпочтительно клетка находится в растении, более предпочтительно в корнях, листьях и/или стеблях растения.
В одном из вариантов осуществления, экспрессия активного NifDK в растительной клетке требует экспрессии NifD, NifK, NifH, NifB, NifE, NifN и, необязательно, NifU, NifS, NifO, NifV, NifY, NifW и/или NifZ.
В другом или дополнительном варианте осуществления, экспрессия активного NifH в растительной клетке требует экспрессии NifH и NifM и, необязательно, NifU и/или NifN.
В одном варианте осуществления, для реконструкции активности нитрогеназы в растительной клетке требуется экспрессия, по меньшей мере, NifD, NifK, NifH, NifB, NifE, NifN и NifM.
Квалифицированному специалисту очевидно, что меньшая подгруппа белков Nif может привести к реконструкции функциональности нитрогеназы в растительной клетке. Насколько известно изобретателям, единственный отчет о переносе гена нитрогеназы в какой-либо фотосинтетический организм описывает введение NifH в хлоропластный геном Chlamydomonas (Cheng et al., 2005). NifH был способен дополнять мутанта биосинтеза хлорофилла, несмотря на то, что белки-предшественники биосинтеза NifH NifM, NifS и NifU не экспрессировались совместно. Это продемонстрировало, что эндогенные эукариотические эквиваленты могут функционально замещать определенные белки Nif. Действительно, недавний отчет, демонстрирующий, что E.coli может восстанавливать функцию нитрогеназы, используя только восемь белков Nif (Wang et al., 2013), подразумевает, что достижение этой функции в растениях может быть менее сложным, чем экспрессия полного набора белков Nif. Хотя изобретатели еще не установили функциональность белков Nif in planta, многообещающим является то, что набор биосинтетических и функциональных белков Nif может быть экспрессирован в среде, потенциально поддерживающей функцию нитрогеназы.
Растения
В контексте данного документа, термин «растение», используемый здесь в качестве существительного, относится к целым растениям и относится к любому члену царства растений, но в качестве прилагательного относится к любому веществу, которое присутствует, получено из, выделено из или связано с растение, например, органы растения (напр., листья, стебли, корни, цветы), отдельные клетки (напр., пыльца), семена, растительные клетки и т.п. Ростки и проросшие семена, из которых выросли корни и побеги, также входят в понятие «растение». В контексте данного документа, термин «части растения» относится к одной или нескольким тканям или органам растения, которые получены из растения и содержат геномную DNA растения. Части растений включают вегетативные структуры (например, листья, стебли), корни, органы/структуры цветков, семена (включая зародыши, семенодоли и семенную оболочку), растительную ткань (например, сосудистую ткань, покровную ткань и тому подобное), клетки и их потомство. В предпочтительном варианте осуществления, часть растения представляет собой семя. В контексте данного документа, термин «растительная клетка» относится к клетке, полученной из растения или в растении, и включает протопласты или другие клетки, полученные из растений, клетки, продуцирующие гаметы, и клетки, которые регенерируют в целые растения. Клетки растений могут представлять собой культуральные клетки. Под «покровной тканью» понимается дифференцированная ткань в растении или полученная из растения («эксплант») или недифференцированная ткань, полученная из незрелых или зрелых зародышей, семян, корней, побегов, плодов, клубней, пыльцы, опухолевой ткани, например, корневого рака и различных форм скоплений растительных клеток в культуре, например, каллусов. Примерными покровными тканями в семенах или из семян являются семенодоли, зародыш и рубчик. Соответственно, изобретение относится к растениям и частям растений и продуктам, их содержащим.
В контексте данного документа, термин «семя» относится к «зрелому семени» растения, которое либо готово для сбора урожая, либо было собрано с растения в поле, или относится к «развивающемуся семени», которое появляется у растения после внесения удобрений до установления состояния покоя семени и перед сбором урожая.
В контексте данного документа, термин «трансгенное растение» относится к растению, которое содержит конструкцию нуклеиновой кислоты, которая не обнаружена в растении дикого типа того же вида, разновидности или сорта. То есть трансгенные растения (трансформированные растения) содержат генетический материал (трансген), который они не содержали до трансформации. Трансген может включать генетические последовательности, полученные или выделенные из растительной клетки, или другой растительной клетки, или нерастительного источника, или синтетической последовательности. Обычно трансген вводят в растение в результате манипуляций человека, например, в результате трансформации, но специалисту в данной области очевидно, что можно использовать любой метод. Генетический материал, предпочтительно, стабильно интегрируется в геном растения, предпочтительно в ядерный геном. Введенный генетический материал может содержать последовательности, которые встречаются в природе у одного и того же вида, но в перегруппированном порядке или при другом расположении элементов, например, антисмысловую последовательность. Растения, содержащие такие последовательности, относятся к «трансгенным растениям».
В предпочтительном варианте осуществления, трансгенные растения являются гомозиготными для каждого введенного гена (трансгена), так что их потомство не сегрегируется по требуемому фенотипу. Трансгенные растения также могут быть гетерозиготными для введенного (-ым) трансгена (-ам),например, в потомстве F1, которое было выращено из гибридного семени. Такие растения могут давать такие преимущества, например, гибридную силу, хорошо известную в данной области.
Трансгенные растения, как определено в контексте настоящего изобретения, дают потомство растений, которые были генетически модифицированы с использованием рекомбинантных методов, где потомство содержит исследуемый трансген. Такое потомство может быть получено в результате самоопыления первичного трансгенного растения или путем скрещивания таких растений с другим растением того же вида. Как правило, это используется для управления продуцированием, по меньшей мере, одного определенного здесь белка в требуемом растении или органе растения. Части трансгенного растения содержат все части и клетки указанных растений, содержащих трансген, например, культивируемые ткани, каллус и протопласты.
Трансгенные растения можно получать методами, известными в данной области, например, которые в описаны в работах A. Slater et al., Plant Biotechnology - The Genetic Manipulation of Plants, Oxford University Press (2003), и P. Christou и H. Klee, Handbook of Plant Biotechnology, John Wiley и Sons (2004).
«Нетрансгенное растение» представляет собой растение, которое не было генетически модифицировано путем введения генетического материала методами рекомбинантной DNA. В контексте данного документа, термин «по сравнению с изогенным растением» или подобные фразы относятся к растению, которое является изогенным по отношению к трансгенному растению, но без исследуемого трансгена. Предпочтительно, чтобы соответствующее нетрансгенное растение принадлежало к тому же сорту или разновидности, что и предшественник исследуемого трансгенного растения, или линии видов-двойников растений, в которой отсутствует конструкция, часто называемая «сегрегант», или растение того же сорта или разновидности, трансформированное конструкцией «пустой вектор», которое может быть нетрансгенным растением. В контексте данного документа, термин «дикий тип» относится к клетке, ткани или растению, которые не были модифицированы согласно изобретению. Клетки, ткань или растения дикого типа можно использовать в качестве контрольных для сравнения уровней экспрессии экзогенной нуклеиновой кислоты или степени и характера модификации признака, с клетками, тканью или растениями, модифицированными, как описано в данном документе.
Трансгенные растения, как определено в контексте настоящего изобретения, дают потомство растений, которые были генетически модифицированы с использованием рекомбинантных методов, где потомство содержит исследуемый трансген. Такое потомство может быть получено в результате самоопыления первичного трансгенного растения или путем скрещивания таких растений с другим растением того же вида. Части трансгенного растения включают все части и клетки указанных растений, содержащие трансген, например, культивируемые ткани, каллус и протопласты.
Растения, предназначенные для использования в практике настоящего изобретения, включают как односемядольные, так и двусемядольные растения. Целевые растения представляют собой, не ограничиваясь, следующие растения: злаки (например, пшеницу, ячмень, рожь, овес, рис, кукурузу, сорго и родственные культуры); виноград; свеклу (сахарную свеклу и кормовую свеклу); семечковые, косточковые и мягкие фрукты (яблоки, груши, сливы, персики, миндаль, вишню, клубнику, малину и жимолость черную); растения из семейства бобовых (фасоль, чечевицу, горох, сою); масличные растения (рапс или другие сорта Brassicas, горчицу, мак, оливки, подсолнечник, сафлор, лен, кокос, касторовое масло, какао-бобы, арахис); растения из семейства огуречных (кабачки, огурцы, дыни); растения из семейства волокнистых (хлопок, лен, конопля, джут); цитрусовые (апельсины, лимоны, грейпфрут, мандарины); овощи (шпинат, салат, спаржа, капуста, морковь, лук, помидоры, картофель, перец); лавровые (авокадо, корица, камфора); или такие растения, как кукуруза, табак, орехи, кофе, сахарный тростник, чай, виноградные лозы, хмель, торф, бананы и растения из натурального каучука, а также декоративные растения (цветы, кусты, широколиственные деревья и вечнозеленые растения, например, хвойные породы). Предпочтительное растение представляет собой злак, более предпочтительно пшеницу, рис, кукурузу, тритикале, овес или ячмень, еще более предпочтительно пшеницу.
В контексте данного документа термин «пшеница» относится к любому виду рода Triticum, включая его предшественников, а также его потомство, полученное путем скрещивания с другими видами. Термин пшеница охватывает «гексаплоидную пшеницу», которая имеет геномную организацию AABBDD, состоящую из 42 хромосом, и «тетраплоидную пшеницу», которая имеет геномную организацию AABB, состоящую из 28 хромосом. Гексаплоидная пшеница охватывает T. aestivum, T. spelta, T. macha, T. compactum, T. sphaerococcum, T. vavilovii и их межвидовые скрещивания. Предпочтительным видом гексаплоидной пшеницы является T. aestivum ssp aestivum (также называемая «пшеница мягкая»). Тетраплоидная пшеница охватывет T. durum (также называемая здесь твердой пшеницей или Triticum turgidum ssp. durum), T. dicoccoides, T. dicoccum, T. polonicum и их межвидовые скрещивания. Кроме того, термин «пшеница» включает потенциальных предков гексаплоида или тетраплоида Triticum sp.,например, T. uartu, T. monococcum или T. boeoticum для генома A, Aegilops speltoides для генома B и T. tauschii (также известную как Aegilops squarrosa или Aegilops tauschii) для генома D. Особенно предпочтительными предшественниками являются предшественники генома A, еще более предпочтительно предшественником генома A является T. monococcum. Сорт пшеницы, для использования в настоящем изобретении, может принадлежать, не ограничиваясь, любым из перечисленных выше видов. Также охватываются растения, которые продуцируют обычными методами с использованием Triticum sp. в качестве родителя при половом скрещивании с видами, не относящимися к Triticum (например, рожь [Secale cereale]), включая, не ограничиваясь, Triticale.
В контексте данного документа термин «ячмень» относится к любому виду рода Hordeum, включая его предшественников, а также его потомство, полученное путем скрещивания с другими видами. Предпочтительно, чтобы это растение принадлежало к виду Hordeum, который культивируется в промышленном масштабе, например, к штамму, или сорту, или разновидности Hordeum vulgare, или было пригодно для промышленного производства зерна.
Способы продуцирования трансгенных растений
Были описаны четыре общих способа прямой доставки гена в клетки: (1) химические способы (Graham et al., 1973); (2) физические способы, например, микроинъекция (Capecchi, 1980); электропорация (см., например, WO 87/06614, US 5 472 869, 5 384 253, WO 92/09696 и WO 93/21335); и генная пушка (см., например, US 4 945 050 и US 5 141 131); (3) вирусные векторы (Clapp, 1993; Lu et al., 1993; Eglitis et al., 1988); и (4) опосредованные рецептором механизмы (Curiel et al., 1992; Wagner et al., 1992).
Способы ускорения, которые можно использовать, включают, например, баллистическую трансфекцию и т.п. Одним из примеров способа доставки трансформирующих молекул нуклеиновой кислоты к растительным клеткам является баллистическая трансфекция. Этот способ был рассмотрен в работе Yang et al., Particle Bombardment Technology for Gene Transfer, Oxford Press, Oxford, England (1994). Небиологические частицы (микрочастицы), которые могут быть покрыты нуклеиновыми кислотами и доставлены в клетки под действием движущей силы. Примерами частиц являются частицы из вольфрама, золота, платины и т.п. Особым преимуществом баллистической трансфекции, помимо того, что она является эффективным средством воспроизводимой трансформации односемядольных, является то, что не требуется ни выделение протопластов ни восприимчивость к инфекции Agrobacterium. Система доставки частиц, подходящая для использования с настоящим изобретением, представляет собой гелиевый ускоритель PDS-1000/He, продаваемый компанией Bio-Rad Laboratories. Для баллистической трансфекции незрелые зародыши или производные клетки-мишени, такие как щиток зародыша или каллус из незрелых зародышей, помещают в твердую культуральную среду.
В другом альтернативном варианте осуществления, пластиды можно стабильно трансформировать. Способ, раскрытый для трансформации пластид у высших растений, включает доставку с помощью генной пушки частиц DNA, содержащих селектируемый маркер, и нацеливание DNA на геном пластиды посредством гомологичной рекомбинации (US 5 451 513, US 5 545 818, US 5 877 402, US 5 932 479 и WO 99/05265).
Перенос, опосредованный Agrobacterium, является широко применяемой системой для введения генов в клетки растений, поскольку DNA можно вводить в ткани целого растения, тем самым обходя необходимость регенерации интактного растения из протопласта. Использование, опосредованных Agrobacterium, интегрирующих векторов растений для введения DNA в клетки растений хорошо известно в данной области (см., например, патенты US 5 177 010, US 5 104 310, US 5 004 863, US 5 159 135). Кроме того, интеграция T-DNA представляет собой относительно точный процесс, приводящий к небольшому числу перестроек. Переносимая область DNA определяется пограничными последовательностями, а промежуточная DNA обычно вставляется в геном растения.
Векторы трансформации Agrobacterium способны реплицироваться в E.coli, а также в Agrobacterium, что позволяет выполнять удобные манипуляции, как описано в работе (Klee et al., Plant DNA Infectious Agents, Hohn и Schell, (издатели), Springer-Verlag, New York, (1985): 179-203). Более того, технические достижения в векторах для опосредованного Agrobacterium переноса генов улучшили расположение генов и сайтов рестрикции в векторах, облегчив конструирование векторов, способных экспрессировать различные гены, кодирующие полипептиды. Описанные векторы имеют удобные мультилинкерные области, фланкированные промотором и сайтом полиаденилирования, для прямой экспрессии встроенных генов, кодирующих полипептид, и подходят для настоящих задач. Кроме того, для трансформаций можно использовать Agrobacterium, содержащую как вооруженные, так и обезвреженные гены Ti. Для тех сортов растений, где трансформация, опосредованная Agrobacterium, эффективна, то она представляет собой способ выбора из-за легкого и определенного характера переноса генов.
Трансгенное растение, полученное с использованием способов трансформации Agrobacterium, обычно содержит один генетический локус на одной хромосоме. Такие трансгенные растения можно назвать гемизиготными по добавленному гену. Более предпочтительным является трансгенное растение, гомизиготное по добавленному структурному гену, т. е. трансгенное растение, которое содержит два добавленных гена, по одному гену в одном и том же локусе на каждой хромосоме из пары хромосом. Гомозиготное трансгенное растение можно получить путем полового скрещивания (самоопыления) независимого сегрегантного трансгенного растения, которое содержит единственный добавленный ген, путем проращивания некоторых полученных семян и анализа полученных растений на наличие исследуемого гена.
Также следует понимать, что два разных трансгенных растения также могут быть скрещены для получения потомства, которое содержит два независимо сегрегирующихся экзогенных гена. Самоопыление соответствующего потомства может дать растения, гомозиготные по обоим экзогенным генам. Также предполагается обратное скрещивание с родительским растением и аут-скрещивание с нетрансгенным растением, а также вегетативное размножение. Описание других способов скрещивания, которые обычно используются для различных признаков и культур, можно найти в работе Fehr, Breeding Methods for Cultivar Development, J. Wilcox (издатель) American Society of Agronomy, Madison Wis. (1987).
Трансформацию протопластов растений можно достигнуть в результате использования способов, основанных на осаждении фосфатом кальция, обработке полиэтиленгликолем, электропорации и их комбинациях. Применение этих систем к различным сортам растений зависит от способности регенерировать этот конкретный штамм растений из протопластов. В работе (Fujimura et al., 1985; Toriyama et al., 1986; Abdullah et al., 1986) описаны иллюстративные способы регенерации злаков из протопластов.
Также можно использовать другие способы трансформации клеток, которые включают, помимо прочего, введение DNA в растения путем прямого переноса DNA в пыльцу, путем прямой инъекции DNA в репродуктивные органы растения или путем прямой инъекции DNA в клетки незрелых эмбрионов с последующим восстановлением влагосодержания высушенных эмбрионов.
Регенерация, развитие и культивирование растений из трансформантов протопласта одного растения или из различных трансформированных эксплантов хорошо известны в данной области (Weissbach et al., Methods for Plant Molecular Biology, Academic Press, San Diego, (1988)). Данный способ регенерации и роста обычно включает этапы отбора трансформированных клеток, культивирования этих индивидуализированных клеток через обычные стадии эмбрионального развития и через стадию укоренения проростков. Аналогичным образом регенерируются трансгенные зародыши и семена. Полученные трансгенные корневые побеги затем высаживают в подходящую среду для выращивания растений, например, почву.
Развитие или регенерация растений, содержащих чужеродный экзогенный ген, хорошо известно в данной области. Предпочтительным является, чтобы регенерированные растения самоопылялись для получения гомозиготных трансгенных растений. В противном случае пыльцу, полученную от регенерированных растений, скрещивают с выращиваемыми из семян растениями агрономически важных направлений. И наоборот, пыльца растений этих агрономически важных направлений используется для опыления регенерированных растений. Трансгенное растение по настоящему изобретению, содержащее требуемую экзогенную нуклеиновую кислоту, культивируют с использованием способов, хорошо известных специалисту в данной области.
Способы трансформации двусемядольных растений, в первую очередь с использованием Agrobacterium tumefaciens, и получения трансгенных растений были опубликованы: для хлопка (US 5 004 863, US 5 159 135, US 5 518,908); сои (US 5 569 834, US 5 416 011); сортов Brassica (5 463 174 США); арахиса (Cheng et al., 1996); и гороха (Grant et al., 1995).
Способы трансформации злаковых растений, например, пшеницы и ячменя, для введения генетической вариации в растение, путем введения экзогенной нуклеиновой кислоты, и для регенерации растений из протопластов или незрелых зародышей растений хорошо известны в данной области, см., например, CA 2 092 588, AU 61781/94, AU 667939, US 6 100 447, WO 97/048814, US 5 589 617, US 6 541 257, а другие способы изложены в WO 99/14314. Предпочтительно трансгенные растения пшеницы или ячменя получают с помощью процедур трансформации, опосредованных Agrobacterium tumefaciens. Векторы, несущие требуемую конструкцию нуклеиновой кислоты, могут быть введены в регенерируемые клетки пшеницы, культивированные тканью растений или эксплантатов или в подходящие растительные системы, например протопласты. Регенерируемые клетки пшеницы предпочтительно происходят из щитка незрелых зародышей, зрелых зародышей, каллуса, полученного из них, или из меристематической ткани.
Для подтверждения присутствия трансгенов в трансгенных клетках и растениях можно выполнить амплификацию с помощью полимеразной цепной реакции (PCR) или анализом саузерн-блот с использованием способов, известных специалистам в данной области. Продукты экспрессии трансгенов можно обнаружить любым из множества способов, в зависимости от природы продукта, которые включают анализ вестерн-блот и ферментный анализ. Одним особенно полезным способом количественной оценки экспрессии белка и обнаружения репликации в различных тканях растений является использование репортерного гена, такого как GUS. После получения трансгенных растений их можно выращивать для получения тканей или частей растений, имеющих требуемый фенотип. Можно собирать ткань растения или части растения и/или собирать семена. Семя может служить источником для выращивания дополнительных растений с тканями или частями, имеющими требуемые характеристики.
«Полимеразная цепная реакция» («PCR») представляет собой реакцию, в которой реплики копий целевого полинуклеотида создаются с использованием «пары праймеров» или «набора праймеров», состоящих из «вышестоящего» и «нижестоящего» праймера, и катализатора полимеризации, например DNA-полимеразы, и обычно термостабильного фермента полимеразы. Способы PCR известны в данной области и описаны, например, в работе «PCR» (M.J. McPherson и S.G Moller (издатели), BIOS Scientific Publishers Ltd, Oxford, (2000)). PCR можно проводить на cDNA, полученной из mRNA с обратной транскрипцией, выделенной из растительных клеток, экспрессирующих полинуклеотид, согласно изобретению. Однако, как правило, будет проще, если PCR проводится на геномной DNA, выделенной из растения.
Праймер представляет собой олигонуклеотидную последовательность, которая способна к гибридизации в сиквенс-специфичном режиме с целевой последовательностью и удлиняться во время PCR. Ампликоны, или продукты PCR, или фрагменты PCR, или продукты амплификации, представляют собой продукты продолжения, которые содержат праймер и вновь синтезированные копии целевых последовательностей. Системы мультиплексной PCR содержат несколько наборов праймеров, которые приводят к одновременному получению более чем одного ампликона. Праймеры могут быть идеально согласованы с целевой последовательностью или могут содержать внутренние несовпадающие основания, что способно привести к введению рестрикционного фермента или сайтов каталитического распознавания/расщепления нуклеиновой кислоты в специфичные целевые последовательности. Праймеры могут также содержать дополнительные последовательности и/или содержать модифицированные или меченые нуклеотиды для облегчения захвата или обнаружения ампликонов. Повторяющиеся циклы тепловой денатурации DNA, отжиг праймеров до их комплементарных последовательностей и продолжение отожженных праймеров с помощью полимеразы приводят к экспоненциальной амплификации целевой последовательности. Термины «целевой» или «целевая последовательность» или матрица относятся к последовательностям нуклеиновых кислот, которые амплифицируются.
Способы прямого секвенирования нуклеотидных последовательностей хорошо известны специалистам в данной области и описаны в работах Ausubel et al. (см. выше) и Sambrook et al. (см. выше). Секвенирование можно осуществлять любым подходящим способом, например, дидезокси-секвенированием, химическим секвенированием или их вариациями. Прямое секвенирование имеет преимущество определения вариации в любой паре оснований конкретной последовательности.
Процессирование растения/зерна
Зерно/семена, согласно изобретению, предпочтительно зерно злаков или другие части растений, согласно изобретению, могут перерабатываться для производства пищевого ингредиента, пищевого или непищевого продукта с использованием любой технологии, известной в данной области.
В одном варианте осуществления, продукт представляет собой муку из цельного зерна, например, мука из цельного зерна ультратонкого помола или муку, состоящую примерно из 100% зерна. Мука из цельного зерна содержит компонент рафинированной муки (рафинированная мука) и фракцию грубого помола (сверхтонкий помол фракции грубого помола).
Рафинированная мука может представлять собой муку, полученную, например, путем измельчения и просеивания очищенного зерна, например, зерна пшеницы или ячменя. Размер частиц рафинированной муки описывается как мука, не менее 98% которой проходит через ткань, имеющую отверстия не больше, чем отверстия в тканой проволочной ткани, обозначенной «212 микрометров (проволока США 70)». Фракция грубого помола содержит, по меньшей мере, отруби и зародыши. Например, зародыш представляет собой растение, находящееся в зачаточном состоянии в ядре зерна. Зародыш содержит липиды, клетчатку, витамины, белок, минералы и фитонутриенты, например, флавоноиды. Отруби содержат несколько слоев клеток и значительное количество липидов, клетчатки, витаминов, белка, минералов и фитонутриентов, например, флавоноидов. Кроме того, фракция грубого помола может содержать алейроновый слой, который также содержит липиды, клетчатку, витамины, белок, минералы и фитонутриенты, например, флавоноиды. Слой алейрона, хотя и считается технически частью эндосперма, демонстрирует многие из тех же характеристик, что и отруби, и поэтому обычно удаляется вместе с отрубями и зародышами в процессе измельчения. Слой алейрона содержит белки, витамины и фитонутриенты, например, феруловую кислоту.
Кроме того, фракция грубого помола может смешиваться с компонентом рафинированной муки. Фракция грубого помола может смешиваться с компонентом рафинированной муки с образованием муки из цельного зерна, получая таким образом муку из цельного зерна с повышенной питательной ценностью, повышенным содержанием клетчатки и повышенной антиоксидантной способностью, по сравнению с рафинированной мукой. Например, фракция грубого помола или мука из цельного зерна может использоваться в различных количествах для замены рафинированной муки или муки из цельного зерна в хлебобулочных изделиях, закусочных и пищевых продуктах. Мука из цельного зерна, согласно изобретению (т.е. муки из цельного зерна ультратонкого помола) также может продаваться непосредственно потребителям для использования в домашней выпечке. В примерном варианте осуществления, гранулометрический состав муки из цельного зерна таков, что 98 мас. % частиц муки из цельного зерна имеют размер менее 212 микрометров.
В дополнительных вариантах осуществления, ферменты, обнаруженные в отрубях и зародышах муки из цельного зерна и/или фракции грубого помола, инактивируются для стабилизации муки из цельного зерна и/или фракции грубого помола. Стабилизация представляет собой процесс, в котором используются пар, тепло, излучение или другие методы обработки для инактивации ферментов, обнаруженных в отрубях и зародышевом слое. Стабилизированная мука сохраняет свои кулинарные свойства и имеет более длительный срок хранения.
В дополнительных вариантах осуществления, мука из цельного зерна, фракция грубого помола или рафинированная мука может быть компонентом (ингредиентом) пищевого продукта и может использоваться для производства пищевого продукта. Например, пищевой продукт может представлять собой: бублик, бисквит, хлеб, булочку, круассан, клецку, английскую булочку, булочку, лаваш, бездрожжевой хлеб, замороженное тесто, тесто, запеченные бобы, буррито, чили, тако, тамале, тортилью, закрытый пирог, готовые к употреблению хлопья, готовые блюда, начинку, блюда, которые можно использовать в микроволновой печи, квадратные шоколадные пирожные с орешками, торт, творожный кекс, кофейный торт, печенье, десерт, выпечку, сладкий рулет, шоколадный батончик, корж, начинку для пирога, детское питание, смесь для выпечки, жидкое тесто, панировку, смесь для подливки, наполнитель для мяса, заменитель мяса, смесь приправ, суповую смесь, подливку, ру, заправку для салата, суп, сметану, лапшу, пасту, лапшу рамен, лапшу чау-мейн, лапшу ло-мейн, добавку в мороженое, мороженое батончик, мороженое рожок, мороженое в вафлях, крекер, гренки, пончик, яичный рулет, выдавленную закуску, фруктовый и зерновой батончик, закуску, пригодную для использования в микроволновой печи, питательный батончик, блин, запеченное в духовке хлебобулочное изделие, крендель , пудинг, продукт на основе гранолы, чипсы для закусок, закуски, закусочную смесь, вафли, основу для пиццы, корм для животных или корм для домашних животных.
В альтернативных вариантах осуществления, мука из цельного зерна, рафинированная мука или фракция грубого помола могут быть компонентом пищевой добавки. Например, пищевая добавка может представлять собой добавляемый к рациону продукт, содержащий один или несколько дополнительных ингредиентов, обычно включая: витамины, минералы, кухонные травы, аминокислоты, ферменты, антиоксиданты, лечебные травы, специи, пробиотики, экстракты, пребиотики и клетчатку. Мука из цельного зерна, рафинированная мука или фракция грубого помола, согласно изобретению, содержит витамины, минералы, аминокислоты, ферменты и клетчатку. Например, фракция грубого помола содержит концентрированное количество пищевых волокон, а также другие важные питательные вещества, например, витамины группы B, селен, хром, марганец, магний и антиоксиданты, которые необходимы для здорового питания. Например, 22 грамма фракции грубого помола, согласно изобретению, обеспечивают 33% от рекомендуемой дневной нормы потребления клетчатки. Пищевая добавка может включать любые известные пищевые ингредиенты, которые будут способствовать общему здоровью человека, примеры включают, не ограничиваясь, витамины, минералы, другие компоненты клетчатки, жирные кислоты, антиоксиданты, аминокислоты, пептиды, белки, лютеин, рибозу, жирные кислоты омега-3 и/или другие пищевые ингредиенты. Добавка может поставляться в следующих формах, не ограничиваясь: растворимые смеси быстрого приготовления для напитков, готовые к употреблению напитки, питательные батончики, вафли, печенье, крекеры, желе, капсулы, жвачку, жевательные таблетки и пилюли. В одном варианте осуществления, добавка с клетчаткой поставляется в форме ароматизированного коктейля или напитка солодового типа, причем этот вариант осуществления может быть особенно привлекательным в качестве добавки с клетчаткой для детей.
В дополнительном варианте осуществления, процесс измельчения может использоваться для получения муки из нескольких видов злаков или крупнозернистой фракции грубого помола из нескольких видов злаков. Например, отруби и зародыши одного вида зерна могут измельчаться и смешиваться с измельченным эндоспермом или мукой из цельного зерна из злаков другого вида. В соответствии с другим вариантом, отруби и зародыши одного вида зерна могут измельчаться и смешиваться с измельченным эндоспермом или мукой из цельного зерна другого вида. Предполагается, что настоящее изобретение охватывает смешивание в любом сочетании одного или нескольких видов отрубей, зародышей, эндосперма и муки из цельного зерна из одного или нескольких видов зерен. Такой подход с использованием нескольких видов зерен может применяться при производстве муки по индивидуальному заказу и извлекать выгоду из качества и питательной ценности нескольких видов злаков для получения одного сорта муки.
Предполагается, что мука из цельного зерна, фракция грубого помола и/или зерновые продукты, согласно изобретению, могут быть получены любым способом измельчения, известным в данной области. Пример осуществления включает измельчение зерна в едином потоке без разделения эндосперма, отрубей и зародышей зерна на отдельные потоки. Чистое и кондиционированное зерно подается в измельчитель для первичного дробления, например, молотковую дробилка, вальцовую дробилку, штифтовую дробилку, дробилку ударного действия, дисковую дробилку, фрикционную дробилку, щелевую дробилку и т.п. После измельчения зерно выгружается и подается на мукопросеиватель. Кроме того, предполагается, что мука из цельного зерна, фракция грубого помола и/или зерновые продукты, согласно изобретению, могут быть модифицированы или улучшены в результате множества других процессов, например: ферментации, инстантизации, экструзии, инкапсуляции, поджаривания, обжаривания и т. п.
Производство солода
Предлагаемый напиток на основе солода, согласно изобретению, включает: алкогольные напитки (включая дистиллированные напитки) и безалкогольные напитки, которые производятся с использованием солода как части или всего исходного материала. Примерами является: пиво, хаппошу (пивной напиток с низким содержанием солода), виски, слабоалкогольные напитки на основе солода (напр., напитки на основе солода, содержащие менее 1% спирта) и безалкогольные напитки.
Соложение представляет собой процесс контролируемого замачивания и прорастания с последующей сушкой зерна, например, зерна ячменя и зерна пшеницы. Такая последовательность важна для синтеза множества ферментов, вызывающих модификацию зерна, и процесса, в ходе которго главным образом деполимеризуются стенки мертвых клеток эндосперма и мобилизуются питательные вещества зерна. В последующем процессе сушки, появляется аромат и цвет в ходе химических реакций карамелизации. Хотя солод в первую очередь и используется для производства напитков, однако он также может использоваться в других промышленных процессах, например, в качестве источника ферментов в хлебопекарной промышленности или в качестве ароматизатора и красителя в пищевой промышленности, например, как солод или как солодовая мука или, косвенно, как солодовый сироп и т. д.
В одном варианте осуществления настоящее изобретение относится к способам получения солодовой композиции. Способ включает также стадии:
(i) предоставление зерна, например, ячменя или пшеницы, согласно изобретению;
(ii) замачивание указанного зерна;
(iii) проращивание замоченных зерен при заранее заданных режимах, и;
(iv) сушку указанных проросших зерен.
Например, солод можно производить любым из способов, описанных в работе Hoseney (Principles of Cereal Science и Technology, Second Edition, 1994: American Association of Cereal Chemists, St. Paul, Minn.). Однако, согласно изобретению, может использоваться любой другой подходящий способ производства солода, например, способы производства специальных солодов, включая, не ограничиваясь, способы обжарки солода.
Солод в основном используется для пивоварения, но также и для производства спиртов, очищенных перегонкой. Пивоварение включает производство сусла, основное и вторичное брожение и доочистку. Сначала солод измельчают, размешивают с водой и нагревают. Во время этого процесса «затирания» ферменты, активированные при соложении, разлагают крахмал ядра до сбраживаемых сахаров. Полученное сусло осветляют, добавляют дрожжи, смесь ферментируют и проводят доочистку.
Обнаружение комплекса нитрогеназы
Обнаружение комплекса нитрогеназы можно проводить любым методом, который позволяет обнаруживать взаимодействие между белковым комплексом NifDK и белком NifH. Способы, подходящие для обнаружения взаимодействия между белковым комплексом NifDK и белком NifH, включают любой способ, известный в данной области, для обнаружения взаимодействия белок-белок, включая совместную иммунопреципитацию, аффинный блоттинг, аффинную адсорбцию, FRET и тому подобное.
Кроме того, обнаружение комплекса нитрогеназы можно проводить путем измерения активности полученного комплекса нитрогеназы.
Способы, подходящие для измерения активности нитрогеназы, включают любой способ, известный в данной области, для обнаружения ферментативного восстановления молекулярного азота до аммиака, при котором электроны переносятся от белка NifH к белковому комплексу NifDK. Например, активность связывания азота можно оценить с помощью анализа восстановления ацетилена. Вкратце, этот способ является косвенным способом, который использует способность комплекса нитрогеназы восстанавливать тройные связанные субстраты. Фермент нитрогеназа восстанавливает ацетилен (C2H2) до этилена (C2H4). Присутствие обоих газов можно количественно определить с помощью газовой хроматографии. Фиксацию азота также можно измерить с помощью анализа выделения водорода. H2 является обязательным побочным продуктом фиксации N2. Косвенную меру активности нитрогеназы можно получить в результате количественного определения концентрации H2 в газовом потоке с использованием проточного датчика H2 или газового хроматографа.
Обнаружение фиксации N2
Фиксацию азота можно оценивать, путем: определения чистого прироста общего N в системе растение-почва (метод баланса N); 2) разделения N растения на фракцию, взятую из почвы, и фракцию, полученную в результате фиксации N2 (отклонение N, естественное содержание 15N, разведение изотипа 15N и уреидные методы) и 3) измерения активности нитрогеназы (анализы восстановления ацетилена и выделения водорода).
ПРИМЕРЫ
Пример 1. Средства и способы
Экспрессия генов в растительной клетке в системе транзиентной экспрессии
Гены экспрессировали в растительных клетках в системе транзиентной экспрессии, по существу, как описано в работе Wood et al. (2009), с различными модификациями, как указано ниже. Растения Nicotiana benthamiana выращивали в вегетационной камере при 23°C при цикле чередования свет:темнота 16: 8 ч с интенсивностью света 90 мкмоль/мин, обеспечиваемой холодными белыми люминесцентными лампами. Бинарные векторы, содержащие кодирующую область, которая должна быть экспрессирована в растительных клетках с помощью сильного конститутивного промотора 35S или усиленного промотора 35S (e35S; Kay et al., 1987), были введены в штамм GV3101 Agrobacterium tumefaciens. Химерный бинарный вектор 35S::p19 для экспрессии вирусного супрессора сайленсинга p19 вводили отдельно в штамм AGL1 A. tumefaciens, как описано в патенте WO2010/057246. Этот вирусный супрессор сайленсинга обычно включали в способ для поддержания экспрессии генов трансгенов, введенных вместе с ним. Рекомбинантные клетки A. tumefaciens выращивали до стационарной фазы при 28°C в питательной среде LB с добавлением 50 мг/л карбенициллина или 50 мг/л канамицина, в соответствии селективным маркерным геном вектора, и добавлением 50 мг/л рифампицина. К культуре добавляли ацетосирингон до конечной концентрации в 100 ммоль/л, а затем культуру инкубировали при 28°C при встряхивании продолжительностью 2,5 часа. Затем бактерии осаждали центрифугированием при 5000 × g в течение 10 мин и при комнатной температуре. Надосадочную жидкость сливали, а осадок повторно суспендировали в растворе, содержащем 10 ммоль MES pH 5,7, 10 ммоль MgCl2 и 100 мкмоль/л ацетосирингона, после чего измеряли OD600. Объем каждой культуры, включая культуру, содержащую конструкцию вирусного супрессора 35S::p19, необходимый для достижения конечной концентрации OD600=0,10, добавляли в неиспользованную пробирку. До окончательного объема доводили инфильтрационным буфером. Затем листья инфильтрировали культуральной смесью, а растения обычно выращивали еще от трех до пяти дней после инфильтрирования, прежде отбора листовых дисков для анализа. Обычно делали контрольную инфильтрацию, которая содержала только конструкцию вирусного супрессора 35S::p19.
Для сверхэкспрессии более чем одного исследуемого гена в комбинации, каждый дополнительный ген вводили отдельно в штамм A. tumefaciens и выращивали, как описано выше. Бактериальные суспензии смешивали таким образом, чтобы каждый штамм бактерий имел конечную концентрацию OD600=0,10. Штамм бактерий, содержащий ген, кодирующий вирусный супрессор сайленсинга 35S::p19, включали во все смеси в одинаковой концентрации. Например, для экспрессии четырех генов при анализе транзиентной экспрессии листьев, включая конструкцию вирусного супрессора, конечный OD600 инфильтрированной смеси составлял 5×0,10=0,50 единиц. Одновременная сверхэкспрессия, по меньшей мере, пяти генов, каждого из отдельных векторов T-DNA в растительных клетках в формате транзиентного анализа была продемонстрирована ранее с использованием Nicotiana benthamiana (Wood et al., 2009).
Конструирование плазмид для экспрессии генов Nif в листьях N. benthamiana
Если не указано иное, плазмиды для транзиентной экспрессии генов в листьях N. benthamiana были сконструированы с использованием метода сборки Golden Gate (Weber et al., 2011). Части DNA в виде отдельных плазмид (Thermo Fisher Scientific, ENSA), каждая из которых содержит промотор 35S CaMV (EC51288), ген, кодирующий первую 51 аминокислоту γ субъединицы F1-ATPase Arabidopsis thaliana (MTP-FAγ51), кодон-оптимизированного растения nifH (EC38011), nifK (EC38015), nifY (EC38019), nifE (EC38016), nifN (EC38024), nifJ (EC38022), nifB (EC38017), nifQ (EC38025), nifF (EC38021), nifU (EC38026), nifS (EC38018), nifV (EC38020), nifW (EC38027), nifZ (EC38029), nifM (EC38023), nifX (EC38028), метку эпитопа HA, кодон-оптимизированного растения (EC38003), и область CaMV последовательности полиаденилирования/терминатора транскрипции (EC41414) были собраны в скелетные векторы (EC47772, EC47742, EC47751, EC47761, EC47781) с использованием рестрикционного клонирования Типа IIS.
Экстракция RNA, синтез и анализ cDNA
Для извлечения RNA из образцов листьев растения, например тех, которые были инфильтрированы Agrobacterium, кусочки листьев размером примерно 2×2 см замораживали в жидком азоте, измельчали до порошка и добавляли 500 мкл буфера Trizol (Thermo Fisher Scientific) на образец. После этого соблюдали инструкции поставщика Trizol, за исключением: повторяли экстракцию хлороформом и растворяли RNA при 37 °C. Экстрагированную RNA обрабатывали RQ1 DNA (Promega) для удаления любой экстрагированной DNA. Затем препараты RNA дополнительно очищали с использованием колонок Plant RNeasy (Qiagen). В ходе выполнения, синтез cDNA осуществляли с использованием обратной транскриптазы Superscript III (Thermo Fisher Scientific) в соответствии с инструкцией поставщика с праймером oligo-dT. Для анализа RT-PCR каждого образца RNA проводили три отдельные реакции синтеза cDNA. Реакционные 20 мкл cDNA разбавляли в 20 раз избыточной водой в нуклеазе. Реакцию qRT-PCR проводили на роторном амплификаторе Rotor-Gene в реальном времени. Для конечного реакционного объема в 20 мкл, 9,6 мкл каждой cDNA добавляли к 10 мкл 2x SensiFAST™ Probe No-ROX Kit (Bioline) и 0,4 мкл прямого и обратного праймеров по 10 мкмоль, каждого. Все реакции qPCR (как для эталонных, так и для специфичных генов) проводят в трижды при следующих режимах цикла: 1 цикл при 95 °C/5 мин, 45 циклов при 95°C/15сек, при 60 °C/15 сек и при 72 °C/20 сек. Флуоресценция измеряется при 72 °C. Затем проводят цикл плавления при температуре, составляющей от 55°C до 99 °C. Контрольные амплификации для конститутивно экспрессированной mRNA N. benthamiana GADPH используют для нормализации экспрессии гена с использованием программы сравнительного количественного анализа в программном пакете амплификатора Rotor-Gene. Значения для каждого набора из трех cDNA, представляющие среднее значение трех анализов, усредняют, что позволяет рассчитать стандартную ошибку среднего (SEM).
Экстракция белка из бактериальных клеток
Белок выделяли из клеток E. coli экстракцией с буфером мочевина/SDS (8 моль/л мочевина, 2% SDS, 100 ммоль трис-HCl pH 8,5, 65 ммоль DTT). Добавляли 300 мкл буфера для экстракции, смесь перемешивали вихревым способом в течение 10 секунд и центрифугировали при 12000 × g в течение 2 минут. Надосадочные жидкости, содержащие экстрагированные белки («общие белки»), хранили при -80°C перед процессированием. Оценку содержания белка проводили с использованием микротитровального анализа белка по методу Брэдфорда (Bio-Rad, California, USA) в соответствии с инструкциями производителя. Для этого, экстрагированные белки из разных образцов разбавляли водой в двух разведениях (1:20, 1:40) и в двух экземплярах, а измерения выполняли при длине волны 595 нм с использованием SpectraMax Plus. Пробу бычьего сывороточного альбумина (BSA) использовали в линейном диапазоне, составляющем от 0,05 мг/мл до примерно 0,5 мг/мл. Концентрацию BSA определяли высокочувствительным аминокислотным анализом Australian Proteomics Analysis Facility (Sydney, Australia). Стандартные кривые, с поправкой на холостую пробу, воспроизводили в двух экземплярах. Для подбора стандартной кривой использовали линейную регрессию.
Извлечение белка из ткани листа
Для анализа количества и свойств специфичных полипептидов, продуцированных в растительных клетках после введения T-DNA, особенно размера полипептидов, как индикатора процессинга в митохондриях, образцы листьев N. benthamiana собирали путем вырезания кусочков листьев, площадью примерно 180 мм2, из инфильтрированных областей через 4 или 5 дней после инфильтрирования, если не указано иное. Листья замораживали в жидком азоте и при процессинге растирали в порошок с помощью ступки и пестика. К каждому образцу порошка добавляли 300 мкл буфера. Буфер содержал 125 ммоль трис-HCl pH 6,8, 4% (мас./об.) додецилсульфата натрия (SDS), 20% (мас./ об.) глицерина, 60 ммоль дитиотреитола (DTT) и 0,002% (мас./об.) бромфенолового синего. Образцы нагревали при 95°C в течение 3 минут перед центрифугированием при 12000 x g в течение 2 минут. Надосадочную жидкость, содержащую экстрагированные полипептиды, называемые здесь образцами «общего белка», удаляли и использовали для анализа вестерн-блот в количестве, составляющем от 10 мкл до 100 мкл, в зависимости от ожидаемого уровня обнаруживаемого полипептида.
Получение общих, нерастворимых и растворимых белковых фракций из ткани листа
Образцы листьев N. benthamiana собирали путем вырезания кусочков листьев, площадью примерно 180 мм2, из инфильтрированных областей через 4 или 5 дней после инфильтрирования. Листья замораживали в жидком азоте и при процессинге растирали в порошок с помощью ступки и пестика.
Для исследования растворимости, собранную ткань листа измельчали в жидком азоте и переносили в микроцентрифужную пробирку, содержащую буфер для экстракции (100 ммоль Трис pH 8,0, 150 ммоль NaCl, 0,25 моль/л маннита, 5% (об./ об.) глицерина, 1% (об./об.) Tween® 20, 1% (мас./об.) PVP, 2 ммоль ТСЕР, 0,2 ммоль PMSF, 10 µмоль/л леупептина). Образец центрифугировали при 20000 x g в течение 5 минут для разделения образца на растворимую (надосадочную) и нерастворимую (осадочную) фракции. Надосадочную жидкость переносили в чистую микроцентрифужную пробирку и снова центрифугировали при 20000 x g в течение 5 минут, и осадок трижды промывали буфером для экстракции. К полученным растворимым и нерастворимым фракциям добавляли буфер Лэммли и подвергали электрофорезу SDS-PAGE с последующим анализом вестерн-блот, как описано в работе Allen et al. (2017).
К каждому измельченному образцу добавляли 300 мкл буфера для определения растворимости в холодной воде. Буфер растворимости для определения растворимости в холодной воде содержал 50 ммоль трис-HCl pH 8,0, 75 ммоль NaCl, 100 ммоль маннита, 2 ммоль DTT, 0,5% (мас./об.) поливинилпирролидона (средняя молярная масса 40 000), 5% (об./об.) глицерина, 0,2 ммоль. PMSF, 10 мкмоль/л леупептина и 0,5% (об./об.) Tween® 20. Образцы центрифугировали в течение 5 минут при 16000 x g при 4 °C. Надосадочную жидкость переносили в чистую пробирку и осадок повторно суспендировали в 300 мкл буфера для определения растворимости в холодной воде. Надосадочную жидкость (образец 1) и повторно суспендированный осадок (образец 2) снова центрифугировали в течение 5 минут при 16000 x g при 4 °C. Из образца 1 отбирали образец надосадочной жидкости, который называется растворимой фракцией. Этот образец смешивали с эквивалентным количеством буфера 4-х SDS. Буфер 4-x SDS содержал 250 ммоль трис-HCl pH 6,8, 8% (мас./об.) SDS, 40% (об./об.) глицерина, 120 ммоль DTT и 0,004% (мас./об.) бромфенолового синего. После второго этапа центрифугирования образец 2 надосадочной жидкости утилизировали. Осадок называется нерастворимой фракцией. Осадок повторно суспендировали в 300 мкл буфера 4-х SDS и добавляли 300 мкл буфера для определения растворимости. Когда растворимые и нерастворимые фракции сравнивали с количеством общего белка, то кусочек листа для образца общего белка измельчали, как описано выше. Однако измельченный образец повторно суспендировали в 300 мкл буфера 4-х SDS и добавляли 300 мкл буфера для определения растворимости. Образцы для общих, нерастворимых и растворимых фракций нагревали при 95°C в течение 3 минут, а затем центрифугировали при 12000 x g в течение 2 минут. Надосадочную жидкость объемом 20 мкл, содержащую экстрагированные полипептиды, загружали в 4-12% гели NuPAGE Bis Tris (Thermo Fisher Scientific) для гель-электрофореза и анализа вестерн-блот.
Для анализа вестерн-блот анаэробно экстрагированных белков экстракции проводили в анаэробной камере (COY Laboratory Products), заполненной атмосферой H2/N2 в сотношении (2-3%/97-98%). Растворы для анаэробной экстракции готовили на линии Шленка в бутылке, снабженной бутилкаучуковой перегородкой, с помощью, по меньшей мере, четырех циклов вакуумирования и продувки N2.
Очистка растений от полипептидов, слитых с эпитопом TwinStrep
Образцы листьев N. benthamiana собирали через пять дней после инфильтрирования Agrobacterium, содержащей исследуемую генетическую конструкцию, или из стабильно трансформированных листьев растений, и обрабатывали следующим образом. Листовой материал массой 15-20 г расщепляли в 100 мл буфера для холодной экстракции в анаэробных условиях (<5 ч/млн. O2) с использованием погружного блендера 6-ю импульсами по пять секунд, сохраняя смесь холодной на льду. Гомогенизированную смесь фильтровали через четыре слоя ткани Mira, а фильтрат (70-80 мл) центрифугировали в течение 30 минут при 3800 g при 4 °C. Надосадочную жидкость декантировали и фильтровали через мембрану PVDF 0,45 мкм для дальнейшего удаления мелких частиц. Фильтрат (60-70 мл) загружали в колонку StreptactinXT (объем слоя 2 мл) со скоростью 2 мл/мин. Колонку промывали 20 мл промывочного буфера перед элюированием полипептидов, содержащих эпитоп TS, с использованием буфера, содержащего 50 ммоль биотина, 50 ммоль Трис pH 8,0 и 75 ммоль NaCl (элюирующий буфер). Собранные фракции под номерами 2-8 по 3 мл каждая дополнительно концентрировали с помощью отделяющей по молекулярной массе 10 кДа мембране (10КДа MWCO, Amersham) путем центрифугирования в течение 30 мин при 3800 × g. Очищенный белковый концентрат мгновенно замораживали в жидком азоте для дальнейшего анализа. Образцы отбирали с каждой стадии процесса очистки для анализа вестерн-блот, проводимого при нормальной атмосфере. Образцы и маркеры молекулярной массы (BenchMark ladder) подвергали электрофорезу в 4-20% гелях NuPage в течение 60 минут при 200 В, используя 20 мкл образца на дорожку. Белки в гелях подвергали блоттингу на мембране PVDF с использованием аппарата iBLOT, а белки, содержащие эпитоп, обнаруживали с помощью антител анти-HA (1: 10000) и антител анти-STREP: HRP (1-этап).
Анализ вестерн-блот
Полипептиды в экстрагированных образцах разделяли электрофорезом в полиакриламидном геле в присутствии додецилсульфата натрия (SDS-PAGE) в 4-12% гелях NuPAGE Bis трис (Thermo Fisher Scientific) при 200 В в течение примерно 1 часа. Разделенные полипептиды из каждого геля переносили на мембрану PVDF, используя сухой аппарат (iBLOT) в соответствии с инструкциями поставщика (Thermo Fisher Scientific), используя трехэтапную 7-минутную программу переноса (1 мин при 20 В, 4 мин при 23 В, и 2 мин при 25 В. После блоттинга гели сохраняли и окрашивали красителем Кумасси (SimplyBlue SafeStain, Thermo Fisher Scientific) в течение ночи, затем промывали водой для визуализации оставшихся белков, чтобы подтвердить перенос полипептидов. Окрашивание красителем Кумасси также обеспечило подтверждение равной нагрузки количества белка на дорожку геля в результате использования уровней очень распространенных белков, например, большой и малой субъединицы Rubisco, в качестве индикатора равной нагрузки белка на дорожку. Мембраны со связанными полипептидами блокировали в течение ночи в буфере TBST, содержащем 5% сухого обезжиренного молока, при 4 °C. Буфер TBST содержал 50 ммоль трис-HCl, pH 7,5, 150 ммоль NaCl и 0,1% (об./об.) Tween®20. Моноклональные антитела анти-НА, продуцированные у мыши, и антитела анти- кроличьей IgG (цельная молекула) пероксидазы, продуцированные у коз, приобретались в компании Sigma-Aldrich. Конъюгат Immun-Star Goat Anti-Mouse (GAM)-HRP приобретали в компании Bio-Rad. Антитело анти-изоцитратдегидрогеназа (IDH), полученное у кролика приобретали в компании Agrisera. Конъюгат StrepMABclassic-HRP на основе антитела приобретали в компании IBA. Антитело анти-GFP было подарком Leila Blackman (Australian National University, Canberra, Australia). Антитела анти-HA, анти-IDH и анти-GFP добавляли разведенными в сотношении 1:5000, конъюгат StrepMABclassic-HRP на основе антител добавляли разведенными в соотношении 1:10000 в TBST с 5% сухого обезжиренного молока, а мембраны инкубировали в растворе в течении от 1 до 2 часов. Затем мембраны промывали TBST 3 раза по 20 минут. В ходе использования конъюгата StrepMABclassic-HRP на основе антитела, антитела обнаруживали на этой стадии с использованием реагента Amersham ECL (GE Healthcare), а мембраны просматривали на устройстве Amersham imager 600 (GE Healthcare). Для анти-HA и анти-GFP конъюгат Mouse-HRP на основе вторичных антител добавляли в соотношении 1:5000 в TBST, содержащий 5% обезжиренного молока, а мембраны инкубировали в течение 1 часа. Для анти-IDH добавляли вторичное антитело анти- кроличьей пероксидазы с разведением в соотношении 1:5000 в TBST, содержащей 5% обезжиренного молока, а мембраны инкубировали в течение 1 часа. Мембраны промывали в TBST 3 х 15 мин. Для обнаружения вторичных антител использовали реагент Amersham ECL, а мембраны просматривали на устройстве Amersham imager.
Обработка белковых экстрактов трипсином перед анализом LC-MS
Образцы белков, используемые для анализа LC-MS, подвергали пробоподготовке вспомогательным фильтрационным веществом (FASP), методом, используемым для расщепления белков в фильтре перед масс-спектрометрическим анализом (Wisniewski et al., 2011). Вкратце, 100 мкл (~ 200 мкг) белка разводили в 100 мкл растора с 8 моль/л мочевины, 100 ммоль Трис-HCl, pH 8,5 (буфер UA) и загружали в центробежный фильтр с отсечкой по молекулярной массе в 10 кДа (MWCO) (Merck Millipore, Australia) и центрифугировали при 20 800 g в течение 15 мин при комнатной температуре (RT). Фильтр с задержанными белками массой > 10 кДа промывали в 200 мкл буфера UA и центрифугировали при 20 800 g в течение 15 мин при комнатной температуре. Для химического уменьшения бисульфидных связей белка в фильтре к 200 мкл раствора добавляли 50 ммоль дитиотреитола и смесь инкубировали при комнатной температуре в течение 50 мин при встряхивании. Фильтр промывали буфером UA двумя объемами по 200 мкл с центрифугированием каждый раз при 20 800 x g в течение 15 мин. Для алкилирования цистеина добавляли 100 мкл раствора йодацетамида (IAM) (50 ммоль IAM в буфере UA), а смесь инкубировали в темноте в течение 30 минут при комнатной температуре перед центрифугированием (20 800 g, 15 минут). Сохраненный белок промывали буфером UA двумя объемами по 200 мкл с центрифугированием (20 800 x g, 15 мин) с последующими двумя последовательными стадиями промывки/центрифугирования в 200 мкл раствора с добавлением 50 ммоль бикарбоната аммония. Раствор трипсина емкостью 200 мкл (степень чистоты реактива для секвенирования Promega, Alexandria, Australia) (20 мкг/мл в 50 ммоль бикарбоната аммония и 1 ммоль CaCl2) загружали на фильтр и инкубировали в течение 1 или 18 часов при 37°C в влажной камере. Триптические пептиды собирали центрифугированием (20 800 x g, 15 мин) с последующей дополнительной промывкой 200 мкл раствора с 50 ммоль бикарбоната аммония. Объединенные фильтраты лиофилизировали и хранили при -20°C.
Жидкостная хроматография с масс-спектрометрией белков
Пептиды, расщепленные трипсином, растворяли в 50 мкл 1% муравьиной кислоты (FA), а аликвоту 4 мкл загружали в аппарат Ekspert nanoLC415 (Eksigent, Dublin, CA, U.S.A.) для хроматографического разделения, напрямую соединенный с аппаратом 6600 TripleTOF MS (SCIEX, Redwood City, CA, USA). Пептиды обессоливали в течение 5 мин в предколонке ChromXP C18 (3 мкм, 120 Å, 10 мм × 0,3 мм) при скорости потока 10 мкл/мин с использованием 0,1% FA и разделяли в ChromXP C18 (3 мкм, 120 Å, 150 мм × 0,3 мм) при скорости потока 5 мкл/мин при 30 °C. Применяли линейный 3-25% градиент растворителя B в течение 68 минут с последующими: 5 мин от 25% B до 35% B; 2 мин от 35% B до 80% B; 3 мин при 80% B, 80-3% B 1 мин; и 8 мин повторного уравновешивания. Растворители представляли собой: (A) 5% DMSO, 0,1% FA, 94,9% воды; (B) 5% DMSO, 0,1% FA, 90% ацетонитрила, 4,9% воды. Параметры прибора: напряжение ионораспыления 5500 В, газовая завеса 25 фунтов на кв. дюйм, GS1 15 фунтов на квадратный дюйм и GS2 15 фунтов на квадратный дюйм, нагреваемая граница раздела 150 °C. Данные были получены в информационно-зависимом режиме сбора данных (IDA), включающем обзорное сканирование по времени пролета (TOF) с последующими 30 MS/MS, каждое с временем накопления 40 мс. Первый этап анализа МС проводили в режиме положительных ионов, диапазон масс m/z составлял от 400 до 1250, а и время накопления 0,25 с. Тандемный масс-спектр получали для ионов-предшественников > 150 импульсов в секунду с зарядовым состоянием 2-5 и динамическим исключением в течение 15 сек и с допуском по массе 100 ч/млн. Спектры получали в диапазоне масс m/z от 100 до 1500 с использованием повторяющейся энергии соударений производителя (CE), основанной на размере и заряде иона-предшественника. Для белков, экстрагированных из E. coli, идентификацию белка осуществляли с использованием программного обеспечения ProteinPilot™ 5.0 (SCIEX) с поисками, проведенными относительно подмножества E. coli в базе данных Uniprot, дополненной специальной базой данных нитрогеназ (Nif+Mit2Nif), включая контрольный белок устойчивости к хлорамфениколу. (CAT/P62577) и базой данных загрязнителей (Common Repository of Adventitious Proteins). Для белков, экстрагированных из N. benthamiana, поиск проводили с использованием подмножества N. benthamiana в базе данных Uniprot, дополненной специальной базой данных нитрогеназы (Nif+Mit2Nif) и базой данных загрязнителей (Common Repository of Adventitious Proteins).
Из идентифицированных пептидов два пептида NifM, а именно DAFAPLAQR (SEQ ID NO:155) и DYLWQQSQQR (SEQ ID NO: 156) были полностью триптическими, не содержали необычных расщеплений и/или модификаций и демонстрировали высокий отклик при МС, который оценивали по пику интенсивности, и были выбраны для селективной регистрации избранных реакций распада нескольких ионов (MRM), чтобы подтвердить обнаружение белков нитрогеназы (NifM) в системе экспрессии E. coli JM109.
Фермент хлорамфеникол-ацетилтрансферазы (CAT; P62577), который обеспечивает устойчивость бактерий к хлорамфениколу, экспрессировали из селектируемого маркерного гена во всех трансформированных клетках E. coli (штамм JM109), содержащих модифицированные или немодифицированные генетические конструкции pMIT2.1. Поэтому этот полипептид был выбран как контрольный для стандартизации уровней экспрессии белка. Для измерения уровня CAT выбирали три триптических пептида (четыре транзиции/пептид) из полипептида CAT, а именно TGYTTVDISQWHR (SEQ ID NO:157), LMNAHPEFR (SEQ ID NO:158) и YYTQGDK (SEQ ID NO:159).
Нацеленная жидкостная хроматография-масс-спектрометрия селективной регистрации избранных реакций распада нескольких ионов (LC-MRM-MS)
Восстановленные и алкилированные триптические пептиды (5 мкл) хроматографически разделяли в колонке Kinetex C18 (2,1 мм x 100 мм, Phenomenex) с использованием линейного 5-45% градиента ацетонитрила в 0,1% муравьиной кислоте в течение 10 мин при скорости потока 400 мкл/мин. Элюент из аппарата UHPLC Shimadzu Nexera направляли для сбора данных и анализа в масс-спектрометр QTRAP 6500 (SCIEX), оборудованный источником ионизации TurboV, работающем в режиме положительных ионов. Параметры МС: напряжение ионораспыления 5500 В; газовая завеса, 35; GS1, 35; GS2, 40; температура источника 500 °С; потенциал декластеризации 70 В; входной потенциал 10 В. Пептиды фрагментировали в ячейке соударений с газообразным азотом и с использованием повторяющейся энергии соударений, основанной на размере и заряде иона-предшественника. Для относительного количественного определения использовали селективную регистрацию избранных реакций распада нескольких ионов (MRM) с 40-секундным окном обнаружения в пределах ожидаемого времени удерживания (RT) и продолжительностью цикла 0,3 секунды. Данные были получены с использованием программного обеспечения Analyst v1.7. Области пиков четырех транзиций MRM интегрировали с использованием Skyline (MacLean, Bioinformatics 2010), при этом для обнаружения все транзиции подлежали совместному элюированию с отношением сигнал-шум (S/N) > 3 и интенсивностью > 1000 импульсов в секунду (имп/сек).
Анализ восстановления ацетилена в E. Coli с использованием системы pMIT2.1
Клетки E. coli штамма JM109 трансформировали плазмидой pMIT2.1 (или одной из исследуемых производных) и контрольной плазмидой pN249, которая придавала устойчивость к антибиотикам: хлорамфениколу и спектиномицину, соответственно, как описано в рабте Temme et al. , 2012. Трансформированные клетки отбирали в ходе роста на среде LB (10 г/л триптона, 5 г/л дрожжевого экстракта, 10 г/л NaCl), содержащей хлорамфеникол (34 мг/л) и спектиномицин (80 мг/л). Трансформированные клетки выращивали в аэробных условиях в течение ночи при 37°C в среде LB с антибиотиками до оптической плотности 1,0 при длине волны 600 нм. Культуры центрифугировали при 10 000 g в течение 1 минуты, а надосадочную жидкость утилизировали. Клетки повторно суспендировали в одном объеме индукционной среды без источников N, содержащей 25 г/л Na2HPO4, 3 г/л KH2PO4, 0,25 г/л MgSO4,7H2O, 1 г/л NaCl, 0,1 г/л CaCl2⋅2H2O, 2,9 мг/л FeCl3, 0,25 мг/л Na2MoO4⋅2H2O и 20 г/л сахарозы (минимальная среда) с добавлением 1,5 мл/л 10 серина, 600 мкл/л 0,5% казаминовых кислот, 5 мг/л биотина и 10 мг/л пара-аминобензойной кислоты (Yang et al., 2018). Среду продували газообразным аргоном в течение 20 минут перед смешиванием с бактериями и антибиотиками. Исходные растворы стерилизовали фильтрованием. Для индукции экспрессии гена Nif в среду добавляли изопропил-β-D-1-тиогалактопиранозид (IPTG; Gold Bio # I2481C25 259) в конечной концентрации 0,1 ммоль, 0,5 ммоль или 1,0 ммоль, если не указано иное, обычно 1,0 ммоль. Суспензии клеток переносили в колбы для культивирования объемом 3,5 см3 и закрывали газонепроницаемыми резиновыми пробками с использованием системы загиба бортика, а подпробковое пространство продували чистым газообразным аргоном в течение 20 минут. Затем суспензии инкубировали при 30°C со встряхиванием при 200 об/мин в течение 5 часов. После этого проводили анализы восстановления ацетилена (ARA) путем инъекции 0,5 см3 чистого C2H2 (газы BOC, разряд прибора; конечная концентрация 10% C2H2 в аргоне) и последующей инкубации в течение 18 часов. Продуцирование этилена в последний моментизмеряли с помощью газовой хроматографии с пламенно-ионизационным детектированием (GC-FID) с использованием прибора Agilent 6890N GC. Образцы из подпробкового пространства (0,5 см3) удаляли и вручную впрыскивали во впускное отверстие с делением/без деления потока 10:1. Параметры прибора: температура на входе и FID 200 °C, средняя скорость носителя He 35 см/сек, температура изотермической печи 120 °C. Использовали колонку RT-Alumina Bond/MAPD (30 м x 0,32 мм x 5 мкм) с предколонкой для частиц 5 м, присоединенной к концу детектора. Аналитические характеристики прибора оценивали путем прогона холостых проб и стандартов. В этих условиях этилен выходил из колонны примерно через 2,3 минуты, а ацетилен - примерно через 3,1 минуты. Эта система GC была способна обнаруживать этилен при очень низком уровне, составляющем 0,00001% атм, с четким разрешением по ацетилену по единственному обнаруживаемому пику в этом формате, и поэтому была чрезвычайно чувствительной.
Система анализа с использованием pMIT2.1 и pN249 дикого типа в штамме JM109 E. coli в качестве позитивной регуляции продуцировала только следовые количества этилена, когда в среду для выращивания не добавляли IPTG, тогда как добавление IPTG в среду для выращивания в концентрации 0,1 ммоль 0,5 ммоль или 1,0 ммоль значительно увеличивало количество продуцируемого этилена. Скорость продуцирования этилена значительно увеличивалась при отборе образцов с 3 часов до 18 часов, а также по мере увеличения концентрации IPTG, что указывало на повышенную активность нитрогеназы с повышенной экспрессией гена Nif. Поэтому в анализах обычно использовали 1,0 ммоль IPTG и отбор образцов проводили через 18 часов.
Трансформация дрожжей и условия культивирования для экспрессии рекомбинантного белка
Трансформацию дрожжевого штамма INVSc1 (Thermo Fisher Scientific) проводили с использованием набора Yeast Transformation Kit (Sigma Aldrich) в соответствии с протоколом производителя. Для векторов, содержащих ген Ura в качестве селектируемого маркера, трансформированные колонии отбирали путем посева смеси для трансформации в чашки с агаром с минимальной средой без урацила (SCMM-U), которая содержала 6,7 г/л дрожжевого азотного основания, 1,92 г/л синтетической выпадающей среды без урацила (Sigma Aldrich), 20 г/л глюкозы и 20 г/л агара. После 2-3 дней инкубации при 30°C отдельные колонии повторно высевали в чашки со свежим агаром SCMM-U. Присутствие генетической конструкции, включающей ген NifD или другой ген Nif, подтверждали с помощью PCR с использованием специфичных для генов праймеров. Единственную колонию, содержащую генетическую конструкцию, инокулировали в жидкую среду SCMM-U (содержащую те же компоненты, что и агар SCMM-U, но без агара), и выращивали при 30°C со встряхиванием в течение 2 дней. Добавляли глицерин до конечной концентрации 20%, а аликвоты хранили при -80°C до дальнейшего использования.
Для экспрессии генов, содержащихся в генетической конструкции, инокулянт из глицеринового исходного материала выращивали в жидкой среде SCMMM-U при 30°C со встряхиванием в течение 2 дней. Клетки собирали из культуры центрифугированием и повторно суспендировали в индукционной среде SCMM-U, которая была идентична жидкой среде SCMM-U, за исключением того, что глюкоза была заменена галактозой 20 г/л до конечной OD600 с 0,4. Культуру для индукции выращивали при 30°C при встряхивании в течение 2 дней, а дрожжевые клетки собирали центрифугированием для экстракции белка и анализа вестерн-блот.
Пример 2. Продуцирование полипептидов Nif в митохондриях растительных клеток путем экспрессии слитых полипептидов MTP-Nif
Авторы изобретения ранее сообщали об успешном продуцировании 16 различных полипептидов Nif в митохондриях растительных клеток путем введения химерных генов, кодирующих трансляционные слияния митохондриальных нацеливающих пептидов (MTP), связанных с N-концами полипептидов Nif (Allen et al., 2017; WO2018/141030). Используемые последовательности MTP включали одну из последовательностей длиной в 77 аминокислот (аминокислоты 1-77 SEQ ID NO:20), полученную из F1-ATPase A. thaliana (At2G33040; Lee et al, 2012) и обозначенную в настоящем документе как MTP-FAγ77, которая с 3-х аминокислотным линкером Gly-Ala-Pro (GAP), связывающим MTP с N-концом полипептидов Nif, обеспечивала продолжение N-конца транслированных полипептидов Nif на 80 аминокислот. Расщепление MPP происходило через 42 аминокислоты, оставляя N-концевое продолжение из 38 аминокислотных остатков, слитых с исследуемым полипептидом Nif, причем 35 остатков происходили от MTP-FAγ77 плюс GAP. Это N-концевое продолжение получило названние FAγ-scar38. Таким образом, метиониновый остаток природной инициации трансляции каждого полипептида Nif был заменен последовательностью scar-38. В ходе этих экспериментов не исследовали нормальную функцию продуцированных полипептидов Nif.
Авторы изобретения стремились сократить последовательность MTP из 77 аминокислот MTP-FAγ77 для использования с полипептидами Nif в растительных клетках, сохраняя при этом функцию MTP. Авторы изобретения исследовали, можно ли удалить 26 аминокислот с С-конца MTP-FAγ77 для образования MTP, обозначенной как MTP-FAγ51 (SEQ ID NO:21). Данная последовательность имела C-концевой GG, добавленный как следствие процедуры клонирования. Авторы изобретения спрогнозировали, что MTP-FAγ51 будет расщепляться MPP после аминокислоты 42, оставляя 9 аминокислот (ISTQVVRNR; SEQ ID NO:22) из MTP-FAγ51 на N-конце процессированного слитого полипептида и связывающий GG как следствие процедуры клонирования. Эта последовательность из 9-ти аминокислот была обозначена как FAγ-scar9 или просто как scar9.
Для исследования функции MTP-FAγ51, по сравнению с более длинной версией, сначала была создана генетическая конструкция, кодирующая этот MTP, слитой с NifH. Модифицированный ген NifH был идентичен гену NifH в pRA10 (кодирующему MTP-FAγ77+GAP::NifH::HA; SEQ ID NO:23), за исключением того, что кодированный полипептид имел MTP-FAγ51, слитой с N-концом NifH, а не MTP-FAγ77. Полипептид все еще включал аминокислоты GAP для целей клонирования. Кодирующая область NifH в обеих конструкциях кодон-оптимизирована для экспрессии в клетках человека на основе нуклеотидной последовательности в pRA10. Обе конструкции включали последовательность, кодирующую метку эпитопа НА на С-конце полипептида Nif, чтобы обеспечить обнаружение и очистку полипептидов с помощью антитела анти-НА. Укороченная конструкция была обозначена как pRA34 (кодирующая MTP-FAγ51+GAP::NifH::HA, SEQ ID NO:24).
Была создана вторая конструкция, обозначенная SN18, кодирующая слитой полипептид NifH, имеющий аминокислотную последовательность, представленную как SEQ ID NO:25, которая включала несколько модификаций, по сравнению с pRA34, направленных на повышение уровней экспрессии. Усиленный промотор 35S (e35S; Kay et al., 1987) использовали для экспрессии слитого белка, а дополнительный N-концевой Met добавляли в качестве инициатора трансляции, 5’-UTR TMV выше кодирующей белок области, а использование кодонов было переключено на использование кодонов A. thaliana. Все эти модификации осуществляли для увеличения уровня экспрессии как на уровне транскрипции, так и на уровне трансляции. Кроме того, аминокислоты GG использовали вместо GAP сразу после MTP. Также была создана третья конструкция, обозначенная SN29, кодирующая слитой полипептид NifH, имеющий аминокислотную последовательность, представленную как SEQ ID NO:26, где полипептид имел метку эпитопа HA сразу после последовательности MTP-FAγ51 (SEQ ID NO:36) и перед последовательностями GG и NifH (MTP-FAγ51::HA::NifH). Обе эти конструкции создавались в соответствии с методами сборки Golden Gate (Weber et al., 2011), которые обеспечивали сборку генетических компонентов в конструкцию по модульному принципу со специфичными компонентами, как описано в работе Engler (2014).
Эти конструкции исследовали на листьях N. benthamiana и сравнивали с более длинной конструкцией pRA10. Белковые экстракты продуцировали из инфильтрированных тканей листа и подвергали электрофорезу SDS-PAGE и анализу вестерн-блот с использованием антитела HA для оценки уровней экспрессии белка и эффективности процессинга MPP. Для контроля размера непроцессированных слитых полипептидов, белковые экстракты из E.coli, экспрессирующие pRA34 и pRA10, воспроизводили на гелях по соседним дорожкам. Бактериальные экстракты давали полипептидные полосы ожидаемого размера для непроцессированного MTP::NifH. Напротив, белковые экстракты из тканей листа N. benthamiana, инфильтрированные этими конструкциями, давали полипептидные полосы меньшего размера, соответствующие размерам, ожидаемым для полипептидов, процессированных MPP. Экспрессия MTP-FAγ51::NifH::HA из pRA34 и SN18 и полипептида MTP-FAγ51::HA::NifH из SN29, каждая давала полосу с меньшей MW, чем MTP-FAγ77+GAP::NifH::HA, в соответствии с разностью в ожидаемом размере между полипептидами из-за укороченной последовательности MTP. Экспрессия из SN18 была, по меньшей мере, такой же сильной, как и из pRA34, хотя обе были сильными. Авторы изобретения пришли к выводу, что укороченный MTP-FAγ51 способен направлять синтетический гибридный полипептид NifH в митохондрии растительных клеток и обеспечивать процессинг в митохондриях с помощью MPP.
Основываясь на успехе с pRA34, SN18 и SN29, кодирующими полипептиды NifH, была исследована более короткая последовательность MTP с другими 15 полипептидами Nif, кодирующими соответствующие версии MTP-FAγ51. Для этого была создана серия генетических конструкций (Таблицы 3 и 4) с использованием методов сборки Golden Gate (Weber et al., 2011). Методы сборки Golden Gate использовали для сборки различных генных элементов, включая промоторы, 5’-UTR, 3’-UTR, N- и C-концевые продолжения и терминаторы. Каждый элемент имел определенные границы, которые допускали модульную сборку и простую замену элементов. Этот способ клонирования с компонентами, описанными в работе Engler et al. (2014), использовали в следующих Примерах для исследования большого количества различных генетических конструкций для продуцирования слитых полипептидов MTP::Nif. Поскольку в методах сборки Golden Gate использовались рестрикционные ферменты типа IIS, которые осуществляли вырезку за пределами их последовательности распознавания, можно было избежать использования сайтов клонирования рестрикционных ферментов в последовательностях сращивания. Это позволило конструировать гены, кодирующие слияния MTP::Nif, без последовательности Gly-Ala-Pro, присутствующей в более ранних конструкциях. Как указано выше, вместо слияний MTP::полипептидов использовали мостик Gly-Gly, который соответствовал методам сборки Golden Gate. Для этой связи выбирали глицин в качестве стандартной аминокислоты из-за того, что он часто встречается в позиции -1 последовательностей MTP. В качестве исключения, в конструкцию для экспрессии слитого полипептида NifK (SN140), эпитоп НА вставляли между MTP-FAγ51 и последовательностью NifK, разделенной мостиком Gly-Gly, и С-концом дикого типа. Это изменение осуществляли, поскольку ранее наблюдалось, что полипептиду NifK для активности требуется С-конец дикого типа без продолжения С-конца (WO2018/141030).
Был создан второй параллельный набор генетических конструкций, которые кодировали полипептиды Nif, локализованные в цитоплазме, а не полипептиды, локализованные в митохондриях. Это было сделано путем замены в конструкциях кодирующей последовательности MTP-FAγ51 на нуклеотидную последовательность, кодирующую аминокислотный мотив 6xHis (SEQ ID NO:27). Мотив 6xHis имел молекулярную массу, аналогичную мотиву FAγ-scar9, полученному в результате опосредованного MPP-расщепления последовательности MTP-FAγ51. Полипептиды, слитые с 6xHis, в остальном идентичны полипептидам MTP-FAγ51::Nif::HA, включая наличие С-концевого эпитопа НА. Таким образом, версии полипептидов 6xHis обеспечивали подходящие регуляторы молекулярной массы для соответствующих MPP-процессированных полипептидов FAγ-scar9::Nif::HA при анализе вестерн-блот. Исключением была контрольная (локализованная в цитоплазме) конструкция для экспрессии NifK (SN72), которая имела N-концевой слитой эпитоп НА без последовательности MTP, а не мотив 6xHis. Генетические конструкции и прогнозируемая молекулярная масса слитых полипептидов для непроцессированных полипептидов MTP-FAγ51::Nif::HA и слитых полипептидов 6xHis перечислены в Таблице 3.
Таблица 3. Плазмиды были сконструированы для исследования эффективности митохондриального нацеливания и растворимости белков полипептидов Nif, экспрессированных в листьях N. benthamiana. Плазмиды, кодирующие локализованные в цитоплазе полипептиды, имели мотив 6xHis вместо последовательности MTP. Предсказанные размеры полипептидов (кДа) были рассчитаны с использованием программного обеспечения VNti.
Последовательности полипептидов NifD и NifS, использованные в этих слияниях, представляли собой последовательности согласно работе Temme et al. (2012). Эти аминокислотные последовательности представлены в SEQ ID NO:18 и SEQ ID NO:19 соответственно. Аминокислотная последовательность NifD из SEQ ID NO:18 отличается от последовательности из 483 аминокислот, представленной как SEQ ID NO:2, шестью аминокислотными заменами в позициях 39, 41, 87, 96, 355 и 483. Аминокислотная последовательность NifS из SEQ ID NO:19 отличается от последовательности из 400 аминокислот, представленной как SEQ ID NO:11, четырьмя аминокислотными заменами в позициях 110, 113, 124 и 290. Все генетические конструкции, обозначенные здесь знаком SN, которые содержат последовательность NifD или последовательность NifS, использовали последовательности согласно работе Temme et al. (2012).
Каждую из конструкций вводили в клетки листа N. benthamiana, а через 5 дней белки экстрагировали из инфильтрированной ткани листа и подвергали анализу вестерн-блот. Образцы из конструкций, экспрессирующих полипептиды 6xHis, и включенные в качестве маркеров молекулярной массы в анализ вестерн-блот для соответствующих MPP-процессированных полипептидов FAγ-scar9::Nif::HA (см. Таблицу 3), подвергали электрофорезу на гелях в соседних дорожках. Для обнаружения полипептидов использовали эпитоп НА, слитой с С-концом каждого полипептида Nif.
Результаты процессирования MPP показаны на Рисунке 1 и сведены в Таблицу 4. При трансляционном слиянии, в виде слияния MTP::Nif MTP-FAγ51, получали расщепленный полипептид MTP::Nif почти для всех полипептидов Nif, но не для всех с одинаковой эффективностью. NifQ был наименее процессированным, при анализе общего белка с помощью вестерн-блот в одном эксперименте были обнаружены только следы процессированной формы, а в другом ничего. Слитые полипептиды NifF, NifM, NifV, NifX, NifY и NifZ только частично процессировались при слиянии с FAγ51, тогда как другие слитые полипептиды Nif, включая NifB, NifE, NifK, NifN, NifS, NifU и NifW, процессировались эффективно, демонстрируя, что эффективность процессинга для разных Nifs может различаться для одного MTP. Слитой полипептид NifD обнаруживали с низкими уровнями, но стабильно находили продукт деградации (см. ниже). Что касается относительных уровней экспрессии, то генетическая конструкция, кодирующая NifY, давала более низкие уровни полипептида, чем другие, за исключением NifD. Считалось, что это связано с более низкими уровнями экспрессии гена NifY, например, более низкой скоростью трансляции по сравнению с другими белками Nif и/или нестабильностью полипептидов. Слияние кодирующей области NifY с MTP, отличной от FAγ51, является одним из подходов к повышению уровня накопления полипептида.
Для некоторых полипептидов Nif наблюдалась разность в количестве локализованного в цитоплазме полипептида (6xHis) по сравнению с полипептидом локализованном в митохондриях. В частности, полипептиды NifB, NifE, NifH, NifU и NifV, нацеленные на митохондрии, накапливались до более высокого уровня, чем соответствующий полипептид, нацеленный на цитоплазму, тогда как уровень накопления других полипептидов Nif был примерно одинаковым между митохондриальными и цитоплазматическими формами. Единственным исключением из этой тенденции был NifN, где полипептид нацеленный в цитоплазму накапливался до большего уровня, чем митохондриальный аналог.
Некоторые полосы полипептидов с более высокой молекулярной массой также наблюдались при анализе вестерн-блот конструкций, кодирующих NifE, NifH, NifB, NifU и NifZ (Рисунок 1). Считалось, что эти полосы могут соответствовать комплексам димеризации, устойчивым к жестким условиям денатурации, используемым при приготовлении образцов. Аналогичные полосы с высокой молекулярной массой наблюдались ранее для различных митохондриально нацеленных белков Nif с использованием разных MTP (Allen et al., 2017).
Анализ вестерн-блот, сравнивающий конструкции pRA10 и SN18, а также конструкции, кодирующие NifH, NifM, NifS и NifU, представлен на Рисунке 2. Образцы для Рисунка 2 включали белки, экстрагированные из парных инфильтратов с или без совместного инфильтрирования pRA25, которая кодирует слитой полипептид MTP-FAγ77::NifK (WO2018/141030), для исследования влияния добавления NifK на экспрессию и/или процессинг MPP. Не наблюдалось никаких различий при добавлении NifK для экспрессии и процессинга NifH, NifM, NifS и NifU.
Эксперименты показали, что аминокислотная последовательность MTP-FAγ51 способна нацеливать все полипептиды Nif в митохондриальный матрикс в растительных клетках и обеспечивать процессинг с помощью MPP, за исключением процессинга полипептида NifQ. Уровни экспрессии полипептида и эффективность процессинга были такими же хорошими, как и для более длинного FAγ MTP. Кроме того, как считается, в некоторых случаях меньшее количество полипептидных полос меньшего размера, которые указывают на продукты деградации, выявлялось с помощью антитела НА на мембранах для блоттинга, например, для pRA34. Авторы изобретения пришли к выводу, что более короткая последовательность MTP может, неожиданно, снизить деградацию MTP::Nif.
Альтернативные MTP
Ряд различных последовательностей MTP исследовали для оценки их эффективности при транслокализации полипептидов Nif в митохондриальный матрикс растительных клеток. Было выбрано несколько MTP разной длины (30-70 аминокислотных остатков). Было предсказано, что они составят разную длину оставшихся аминокислотных остатков («последовательность scar» или просто «scar»), слитых на N-концах полипептидов Nif после расщепления с помощью MPP (Таблица 5). Последовательности scar имели в длину от 0 до 36 аминокислотных остатков. С помощью методов сборки Golden Gate были собраны 17 различных генетических конструкций с использованием комбинаций этих MTP с несколькими Nif для экспрессии в растительных клетках, особенно для экспрессии слитых полипептидов NifD, поскольку NifD был наиболее сложным для экспрессии полипептидов Nif (WO2018/141030). Промоторы, 5’и 3’ UTR и терминаторы были идентичны для этих конструкций.
Культуры A. tumefaciens, содержащие эти конструкции, каждая из которых смешана с конструкцией, продуцирующей белок супрессора сайленсинга P19, индивидуально вводили в листья N. benthamiana, как описано в Примере 1, а белковые экстракты продуцировали через 5 дней после инфильтрирования. Анализ белковых экстрактов проводили электрофорезом SDS-PAGE и анализом вестерн-блот. Для инфильтрирования с помощью конструкции MTP:NifD, была совместно инфильтрирована конструкция SN46 (pSu9::NifK), поскольку было показано, что совместная экспрессия NifK без С-концевого продолжения увеличивает содержание NifD (WO2018/141030).
Были исследованы две версии CPN60 MTP слитого с NifD. В одной версии MTP был слит таким образом, что линкер Gly-Gly был помещен между CPN60 MTP (SEQ ID NO:28) и NifD (SN11). В каждом случае, если он имеет место, линкер Gly-Gly вставляли методом сборки Golden Gate, но его можно было рассматривать как часть последовательности MTP. В другом варианте (SN4), CPN60 MTP (SEQ ID NO:29) была слита непосредственно с первым метионином полипептида NifD. Поскольку было предсказано, что CPN60 расщепляется сразу после C-концевого тирозина в его аминокислотной последовательности, то эта конструкция теоретически будет продуцировать полипептид NifD с N-концом дикого типа, т. е. без «scar», тогда как конструкция SN11, как предполагалось, оставит продолжение Gly-Gly после расщепления слитого MTP (GlyGly)::NifD. Удивительно, но эти очень похожие конструкции дали разные результаты, о чем свидетельствует анализ вестерн-блот: Конструкция SN11 дала полосу полипептида с размером, ожидаемым для непроцессированного CPN60 (GlyGly)::NifD, тогда как SN4 дала полосы, соответствующие как процессированным, так и непроцессированным полипептидам, причем присутствовало больше непроцессированного полипептида, чем процессированного полипептида. Кроме того, когда белок при инфильтрировании с SN4 сравнили в ходе анализа вестерн-блот с белком, экстрагированным из параллельного инфильтрирования pRA24+pSN46 (FAγ77+GAP::NifD::HA+Su9::NifK), стало очевидно, что конструкция SN4 продуцировала значительно менее правильно процессированный полипептид, чем конструкция pRA24. Следовательно, хотя CPN60 MTP был способен нацеливаться на слитой полипептид и допускал матричный процессинг для процессирования полипептида NifD дикого типа, однако уровень экспрессии и эффективность процессинга были низкими (US2016/0304842). Для SN11 связь Gly-Gly между CPN60 и NifD могла препятствовать процессингу MTP.
Несколько MTP, происходящих из полипептидов супероксид-дисмутазы (SOD), также исследовались как одиночные или как тандемные MTP, либо с включением Ile и Gln на С-конце перед связыванием Gly-Gly, либо без него. Полипептиды не были обнаружены в ходе анализа вестерн-блот для версий, содержащих SOD MTP (SN15, SEQ ID NO:32 и SN16, SEQ ID NO:33), которые не содержали остатков Ile и Gln, тогда как версии, содержащие SOD MTP, которые сохранили остатки Ile и Gln (SN12, SEQ ID NO:30 и SN13, SEQ ID NO:31) действительно продуцируют обнаруживаемые полипептиды, хотя, как оказалось, они не процессировались MPP. Напротив, другой исследованный MTP, L29 (SN17, SEQ ID NO:34), показал сильные полипептидные сигналы при слиянии с NifD. Из-за небольшой разницы в размере между процессированными и непроцессированными формами с этим MTP, потребуются дополнительные эксперименты для определения эффективности процессинга. Ожидается, что MTP L29 дает расщепленный полипептид Nif с высокой эффективностью. Авторы изобретения также исследовали MTP CoxIV с меткой(Burén et al., 2017)twin strep, слитой на С-конце MTP, но выше связи Gly-Gly (SN19, SEQ ID NO:37). Данный MTP при слиянии с NifD также давал сильные сигналы в ходе анализа вестерн-блот, размер которых соответствовал процессингу митохондриального матрикса.
Таблица 4. Обобщенный уровень процессинга MPP полипептидов MTP::Nif, растворимости в митохондриях растений и функциональности scar9-Nif при бактериальном анализе. Да (p) указывает на частичный (менее 50%) процессинг MPP. Показатели растворимости процессированных NIF: -, нерастворимые или малорастворимые; +, частично растворимые, ++ частично или полностью растворимые. Nt= не исследовалось. Функциональное исследование проводилось на штамме JM109 E. coli с использованием векторной системы MIT2.1.
Таблица 5. Подробная информация о MTP, используемая для исследования в растениях с помощью методов сборки Golden Gate. кДа F/ P: полный размер непроцессированного MTP/процессированный размер MTP в кДа. Scar=спрогнозированная аминокислотная последовательность остающегося N-концевого продолжения после процессинга MPP.
1,19
(Prasad and Stewart, 1992)
0,13
0
(Huang et al., 2009)
0,37
0,37
(At3g10920);(Marques et al., 2014)
0,13
(Marques et al., 2014)
0,13
(Huang et al., 2009)
1,36
(SEQ ID NO:40)
0,38
(ABM97483)
(Burén et al., 2017)
3,64
(SEQ ID NO:43)
(ABM97483)
(не опубликовано)
1,84
Пример 3. Растворимость слитых полипептидов Nif в митохондриях растения
Растворимость белковых компонентов нитрогеназы в митохондриальном матриксе считается предпосылкой для функциональной реконструкции нитрогеназы в митохондриях растительных клеток. Хотя полипептиды Nif, такие как NifD, растворимы в азотфиксирующих бактериях, не было известно, будет ли экспрессия слитых синтетических полипептидов MTP::NifD в растительных клетках образовывать растворимые полипептиды, которые могут связываться с другими компонентами Nif, особенно в митохондриальном матриксе. Нерастворимость может быть следствием многих факторов, включая образование агрегатов и ассоциацию с клеточными мембранами, и, вероятно, препятствовать функционированию.
Поэтому изобретатели оценили растворимость полипептидов MTP-FAγ51::Nif::HA и несколько других после экспрессии генетических конструкций (см. Таблицу 4) в клетках листьев N. benthamiana. Белковые экстракты для растворимых и нерастворимых фракций приготовляли, как описано в Примере 1, а также нефракционированные образцы «общего белка», которые содержали как растворимые, так и нерастворимые белки. Буфер для приготовления растворимой фракции содержал неионный детергент Tween®20, который добавляли для лизирования мембран и высвобождения белков митохондриального матрикса. С низкой вероятностью считалось, что этот мягкий неионный детергент денатурирует полипептиды Nif. Напротив, белки нерастворимой фракции переводили в растворимую форму перед электрофорезом в геле с помощью буфера, содержащего относительно высокую концентрацию SDS, сильного анионного детергента, который, как известно, эффективно денатурирует белки, и обработки при высокой температуре. Затем образцы подвергали электрофорезу в геле и анализу вестерн-блот с использованием антитела анти-НА для обнаружения полипептидов на мембранах для блоттинга.
Проводили несколько наблюдений, чтобы исследовать, правильно ли способ различает растворимые и нерастворимые белки. Окрашивание красителем Кумасси остальных полипептидов на гелях после переноса показало, что рибулозо-1,5-бифосфаткарбоксилаза/оксигеназа, как и ожидалось, присутствовала в растворимой фракции. В нерастворимой фракции были обнаружены только следовые количества рибулозо-1,5-бифосфаткарбоксилазы/оксигеназы.. Вестерн-блоты также анализировали с использованием антитела изоцитратдегидрогеназы (IDH). IDH представляет собой оксидоредуктазу, которая участвует в цикле лимонной кислоты и, как известно, растворима и локализуется в митохондриальном матриксе. Анализ вестерн-блот показал присутствие IDH в растворимой фракции, что указывает на то, что митохондрии были успешно лизированы и белки митохондриального матрикса, которые, как ожидается, должны быть растворимы, действительно присутствовали в растворимой фракции. Эти наблюдения показали, что растворимые белки были успешно экстрагированы и фракционированы в растворимый образец использованными способами.
Затем этот способ был применен к слитым полипептидам Nif; репрезентативный анализ вестерн-блот приведен на Рисунке 3, а результаты обобщены в Таблице 4. Содержание MPP-процессированных полипептидов Nif в растворимой фракции варьировало для разных pFAγ51::Nif::HA. Следующие MPP-процессированные полипептиды из продуктов трансляции слитого полипептида MTP-FAγ51::Nif::HA оказались растворимыми или в основном растворимыми в митохондриях: NifF, NifM и NifU. Другие слитые полипептиды NifN, NifQ, NifS, NifW, NifY и NifZ были частично растворимыми/частично нерастворимыми. Следующие вещества оказались нерастворимыми или мало растворимыми: NifB, NifD, NifE, NifH, NifJ, NifK (с N-концом метки эпитопа HA последовательности NifK), NifV и NifX. Примечательно, что pFAγ51::NifQ::HA дает слабую полосу, примерно равную размеру правильно процессированной формы в растворимой фракции, которая не обнаруживалась в дорожке общего белка. Особенно важно, что каждый из полипептидов MTP-FAγ51::NifD::HA (из SN10), MTP-FAγ51::NifE::HA (из SN38) и MTP-FAγ51::HA::NifK (из SN140), при экспрессии в виде отдельного полипептида были практически нерастворимыми, а митохондриально-растворимые формы этих полипептидов не были обнаружены, даже несмотря на то, что значительные количества полипептидов накапливались в клетках листа N. benthamiana. Для слитых полипептидов NifH MTP-FAγ77::NifH (из SN150) при экспрессии в виде отдельного полипептида был по существу нерастворимым, тогда как только небольшое количество MTP-CoxIV::twin strep::NifH (из SN42), при экспрессии в виде отдельного полипептида, было растворимо. Более того, полипептид MTP-FAγ51::NifD (из SN10) при совместной экспрессии с полипептидом MTP-Su9::NifK из SN46 также был практически нерастворим. Был сделан вывод, что для функции нитрогеназы каждый из этих четырех основных полипептидов был проблематичным с точки зрения растворимости при экспрессии для импорта в митохондриальный матрикс.
Для оценки влияния кислорода воздуха на растворимость белка Nif, те же 16 белков pFAγ51::Nif::HA выделяли из инфильтрированных растений в анаэробных условиях, как описано в Примере 1, и подвергали анализу вестерн-блот, как и ранее. Было замечено, что анаэробные условия во время экстракции белка существенно не меняли растворимость слитых полипептидов Nif. Был сделан вывод, что наблюдаемая нерастворимость некоторых полипептидов Nif не связана с воздействием кислорода, хотя многие полипептиды Nif чувствительны к кислороду.
Дальнейший анализ вестерн-блот показал: Полипептид MTP-FAγ51::NifB::HA (продуцированный из SN192) был нерастворим, а в растворимой фракции не было обнаружено полосы. NifB также важен для функции нитрогеназы. Полипептид MTP-FAγ51::NifF::HA (SN138) был почти полностью растворим для обоих полипептидов до и после MPP-процессинга, а на мембранах для блоттинга были обнаружены две полосы, которые, как предполагалось, представляли собой MPP-процессированные и непроцессированные формы. Полипептид MTP-FAγ51::NifJ::HA (SN139) был практически нерастворим, а в растворимой фракции была обнаружена только очень слабая полоса. Полипептид MTP-FAγ51::NifM::HA (SN30) был в основном растворим после MPP-процессинга. Для MTP-FAγ51::NifS::HA (SN31) на мембранах для блоттинга наблюдались две полосы, которые, как предполагалось, представляли MPP-процессированные и непроцессированные полипептиды. Оба были частично растворимы. Полипептид MTP-FAγ51::NifV::HA (SN142) был практически нерастворим, а в растворимой фракции была обнаружена только очень слабая полоса. Полипептид MTP-FAγ51::NifX::HA (SN144) был частично растворим после MPP-процессинга. Полипептид MTP-FAγ51::NifY::HA (SN145) был в основном растворим, хотя в этом эксперименте экспрессировался только на низком уровне. Полипептид MTP-FAγ51::NifZ::HA (SN146) частично находился в растворимой фракции, частично в нерастворимой. В этом эксперименте и рибулозо-1,5-бифосфаткарбоксилазы/оксигеназа, и IDH присутствовали в «общем белке» и растворимых фракциях и по существу отсутствовали в нерастворимых фракциях, показывая, что способ, использованный для фракционирования, был эффективным и что растворимые белки действительно экстрагировались.
В попытке определить причину этих проблем с растворимостью были созданы генетические конструкции, кодирующие версии слитых полипептидов NifD, NifH и NifK, в которых отсутствовала N-концевая последовательность MTP. Было предсказано, что эти полипептиды локализуются в цитоплазме растительных клеток, а не в митохондриях. Конструкции, кодирующие NifD (SN33), NifH (SN71), NifK (SN72), получали методами сборки Golden Gate, где каждый полипептид имеющий только Gly-Gly-связанную метку эпитопа НА, сливался с N-концом последовательности Nif. Например, SN33 кодировал слитой полипептид HA:NifD без С-концевой метки эпитопа HA, поэтому, по существу, N-концевую последовательность MTP-FAγ51 заменяли последовательностью эпитопа HA. Каждую из этих трех конструкций вводили отдельно в клетки N. benthamiana с помощью A. tumefaciens, а анализ вестерн-блот полипептидов, проводили на фракциях растворимых и нерастворимых белков. Анализ вестерн-блот показал, что каждый из полипептидов был практически полностью растворим в растительных клетках. Был сделан вывод, что проблемы растворимости слитых полипептидов NifD, NifH и NifK при слиянии с последовательностью MTP каким-то образом связаны с нацеливанием полипептидов Nif на митохондрии растений.
Пример 4. Функциональные исследования слитых полипептидов Nif после расщепления MTP
В Примере 2 описано продуцирование слитых полипептидов Nif в клетках листа N. benthamiana, а также доставка и процессинг слитых полипептидов в митохондриях. Слитые полипептиды были разработаны для слияния в рамке считывания MTP, добавленного к N-концу полипептидов Nif, и добавленной метки эпитопа, а иногда как N-концевое продолжение, но чаще всего как C-концевое продолжение. Хотя моделирование укладывания и ассоциации белка предсказывало, что большая часть N-концевых и C-концевых продолжений не должна прекращать образование комплекса и функцию нитрогеназы, однако изобретатели хотели исследовать, могут ли эти продолжения влиять на функцию слитых полипептидов относительно нативных полипептидов Nif. Для этого была создана бактериальная система для исследования функции нитрогеназы с использованием производных вектора pMITv2.1 (Smanski et al., 2014; упоминается здесь как pMIT2.1 или MIT2.1). Все гены дикого типа, необходимые для активности нитрогеназы, содержались в одном бактериальном экспрессионном векторе pMIT2.1, где экспрессия генов контролировалась с помощью системы индуцибельный промотор/Т7-RNA-полимераза из второй плазмиды pN249. В ходе экспрессии в E. coli был продуцирован полный набор бактериальных полипептидов Nif дикого типа, которые вместе образовали ферментный комплекс нитрогеназы, активность которого можно было оценить по продуцированию этилена из ацетилена (анализ восстановления ацетилена, ARA), т. е. измерение активности нитрогеназы de facto.
Эта система позволяла анализировать каждый модифицированный полипептид индивидуально в E. coli путем добавления к другому комплексу нитрогеназы дикого типа. Это делали путем замены гена Nif в pMIT2.1, кодирующего полипептид Nif дикого типа, на соответствующий модифицированный ген Nif, кодирующий слитой полипептид Nif для исследования. Комбинации модификаций двух или более полипептидов Nif также могут исследоваться в этой системе. Однако вектор pMIT2.1 был очень большим (22 946 п.о.), что делало его громоздким для включения генетических модификаций. Чтобы сделать векторную систему pMIT2.1 более работоспособной, плазмиду MIT2.1 сначала делили на две половины с помощью PCR. Первую половину, содержащую гены NifHDKYENJ, амплифицировали с использованием праймеров, включающих сайты рестрикционного фермента SbfI на каждом конце, а именно MIT_V2.1_SbfInifH_FW2 5’ -AACCTGCAGGTGACGTCTAAGAAAAGGAATATTCAGCAAT-3’ (SEQ ID NO:45) и MIT_V2.1_SbfInifJ_RV2 5’-AACCTGCAGGGCTAACTAACTAACCACGGACAAA AAACC-3’ (SEQ ID NO:46) и лигировали в реципиентный вектор pCR Blunt II TOPO (Thermo Fisher Scientific), образуя вектор, обозначенный здесь как pTopoH-J. Вторую половину кластера генов Nif, содержащую гены NifBQFUSVWZM, амплифицировали с использованием праймеров, которые также включали сайты рестрикционного фермента SbfI на каждом конце, а именно MIT_V2.1_SbfInifB_FW 5’-AACCTGCAGGTACTCTAACCCCATCGGCCGTCTTA-3’ (SEQ ID NO:47), и MIT_V2.1_SbfIori_RV 5’-AACCTGCAGGTACGTAGCAATCAACTCACTGGCTC-3’ (SEQ ID NO:48). Этот продукт PCR расщепляли SbfI и подвергали самолигированию с образованием самореплицирующегося вектора, обозначенного здесь pB-ori. Для преобразования pMIT2.1 и его производных, как pTopoH-J, так и pB-ori, или производного с модификацией, SbfI расщепляли, а две половины кластера генов Nif лигировали вместе.
Как описано в Примере 2, аминокислотная последовательность MTP-FAγ51 была расщеплена в митохондриях растения с образованием 9 аминокислотных остатков (FAγ-scar9; SEQ ID NO:22) и промежуточного линкера Gly-Gly, в случае конструкций SN, слитых с N-концом полипептида Nif процессированных слитых полипептидов Nif. Чтобы исследовать каждый из слитых полипептидов на их функцию в другом комплексе нитрогеназы дикого типа, фрагмент DNA, кодирующий 9 аминокислот, за исключением замены N-концевого остатка Ile на Met для инициации трансляции (MSTQVVRNR, SEQ ID NO:49, обозначенного mscar9) вставляли непосредственно перед кодоном инициирующим трансляцию каждого гена Nif в pMIT2.1 с использованием описанной выше стратегии. Исключением был NifX, поскольку pMIT2.1 не включает NifX, и поэтому модифицированный NifX не мог быть исследован в этой системе. Для каждой конструкции фрагмент DNA конструировали таким образом, чтобы при слиянии в рамке считывания непосредственно перед инициирующим кодоном гена, кодирующего любой из полипептидов Nif, химерный ген кодировал трансляционное слияние с выбранным полипептидом Nif. Ожидалось, что инициатор трансляции Met будет удален посттрансляционно в E. coli, потому что серин во второй позиции, как известно, способствует запуску удаления Met ферментом MAP (Hirel 1989, Xiao 2010). Если бы это произошло, то результирующее N-концевое продолжения имело бы 8 аминокислотных остатков. Удаление исходного остатка Met подтверждали тщательным сканированием продуцирующих ионов в ходе селективной регистрации избранных реакций распада целевых ионов полутриптического пептида STQVVR (SEQ ID NO:50), с помощью жидкостной хроматографии с тандемной масс-спектрометрией Q-TRAP (см. ниже).
Для бактериальных полипептидов Nif дикого типа, в которых инициирующий трансляцию остаток Met был удален в бактериях посттрансляционно, длина N-концевого продолжения каждого белка Nif составляла 9 аминокислот с последовательностью STQVVRNRM (SEQ ID NO:51) слитой с остатком Nif, где концевой Met представлял собой аминокислоту инициации трансляции полипептида Nif.
В качестве примера модификации pMIT2.1 и ее исследования, в данном случае для введения трансляционного слияния девяти аминокислот пептида mscar9 MSTQVVRNR (SEQ ID NO:49) с N-концом полипептидов Nif, нуклеотидную последовательность, кодирующую эти аминокислоты добавляли к 5’ концу прямого праймера, который создавал гибрид с 5’ концом кодирующей последовательности для каждого гена Nif. Для каждого модифицируемого гена Nif конструировали обратный праймер рядом с 5’ концом конкретного гена Nif. Амплифицированный продукт PCR лигировали с использованием реакции циклического лигирования (LCR; de Kok et al., 2014), после чего другую половину pMIT2.1, которая не была модифицирована, повторно лигировали с модифицированной половиной после расщепления SbfI. Например, для осуществления трансляционного слияния MSTQVVRNR (SEQ ID NO:49) с N-концом NifB, праймеры 5’- ATGTCAACTCAAGTGGTGCGTAACCGCATGACCTCTTGTTCGTCGTT-3’ (SEQ ID NO:52) и 5’-TTTAGCCCTCCTATGATTGATTTGATGTATTACAGAGAGG-3’ (SEQ ID NO:53) использовали в PCR с pB-ori в качестве матрицы с получением продукта, имеющего 11 565 п. о. Фрагмент PCR лигировали с помощью LCR с мостиковым олиго 5’-GGTTACGCACCACTTGAGTTGACATTTTAGCCCTCCTATGATTGATTTGATG-3’ (SEQ ID NO:54), используя способ, описанный в работе de Kok et al. (2014) и использовали для трансформации E. coli DH5α. Полученную конструкцию pB-ori_scar9B расщепляли с помощью SbfI и лигировали фрагментом SbfI из pTopoH-J, содержащим немодифицированные гены NifHDKYENJ, с получением модифицированного вектора pMIT2.1, кодирующего слитой полипептид, имеющий N-концевое продолжение, добавленное к NifB, обозначенный в настоящем документе как pSO006. Нуклеотидные последовательности полученных модифицированных генетических конструкций были подтверждены секвенированием модифицированной половины, будь то половина pTopoH-J или половина pB-ori.
Каждую генетическую конструкцию вводили в штамм JM109 E. coli, содержащий pN249, а культуры клеток, трансформированных обоими векторами, выращивали, как описано в Примере 1. В качестве негативной регуляции использовали pB-ori, лишенный 7 из 16 генов Nif. Измененный pMIT2.1 без NifM, обозначенный как ΔNifM, включали в эксперименты (см. Lei et al., 1999; Howard et al., 1986). Трансформированные клетки исследовали на продуцирование этилена в анализах восстановления ацетилена после индукции экспрессии гена с помощью IPTG. Результаты, обобщенные в Таблицах 4 и 5, показывают процентное содержание в JM109, рассчитанное как активность восстановления ацетилена в штамме JM109 E. coli, содержащем модифицированный pMIT2.1, по сравнению с JM109, содержащим немодифицированный pMIT2.1. Контроль за немодифицированной pMIT2.1 показывал положительное продуцирование этилена. Эти анализы показали, что добавление продолжения mscar9 из 9-ти аминокислот к N-концу NifB немного увеличило функцию нитрогеназы в отношении уровня продуцирования этилена, наблюдаемого у немодифицированного pMIT2.1.
Аналогичным образом, оставшиеся 15 Nif также имели продолжение в 9 аминокислот на своих соответствующих N-концах с полной активностью для NifH, NifJ, NifQ и NifF, но с некоторым снижением активности для других Nif. В первом эксперименте 9 продолжений аминокислот на N-концах NifH, NifD, NifK, NifE и NifN дали уровни активности восстановления ацетилена, которые составили 100%, 50%, 70%, 30% и 50% по сравнению с уровнями, полученными из немодифицированного pMIT2.1, соответственно. Другие полипептиды Nif, а именно NifJ, NifY, NifQ, NifF, NifU, NifS, NifV, NifW, NifZ и NifM, показали уровень активности, составляющий 200%, 60%, 100%, 100%, 80%, 50%, 90%, 30%, 60% и 10% , соответственно, по сравнению с немодифицированным pMIT2.1 (Таблица 4).
Эксперимент повторяли несколько раз, а среднее значение (n=от 2 до 6) приведено в Таблице 6. Это функциональное исследование отдельных полипептидов scar9::Nif в E. coli показало, что активность сохранялась у всех 16 слитых полипептидов Nif, хотя уровни активности для разных Nif значительно варьировались. Примечательно, что scar9::NifJ имел в три раза большую активность по сравнению с позитивной регуляцией, а scar9::NifQ, scar9::NifH, scar9::NifB и scar9::NifF имели значительно большую активность ARA по сравнению с соответствующими полипептидами Nif дикого типа, но демонстрировали примерно 130-150% активности по сравнению с немодифицированным pMIT2.1 и, следовательно, меньше, чем увеличение, наблюдаемое с scar9::NifJ. Напротив, scar9::NifM сохранял только примерно 10% активности по сравнению с NifM дикого типа.
Учитывая высокую активность scar9-NifJ (pSO028) в системе pMIT2.1, которая в 2-3 раза более активна по сравнению с немодифицированной регуляцией, было дополнительно исследовано влияние модификации NifJ. Вся область NifJ из pMIT2.1 была удалена, давая ΔNifJ-MIT2.1 (pSO014). Анализы восстановления ацетилена с pSO014 показали, что его активность аналогична pMIT2.1, что указывает на то, что NifJ был избыточным в системе анализа ARA в штамме JM109. Следовательно, повышенная активность с scar9-NifJ (pSO028) в системе pMIT2.1 могла быть связана с эффектом дозировки гена.
Эксперименты, описанные в Примерах 2-4, привели авторов изобретения к выводу, что содержание, процессинг MPP и растворимость 16 различных полипептидов MTP::Nif варьировались, несмотря на использование одного и того же MTP и промотора для каждой экспрессирующей конструкции. Однако все слитые полипептиды Nif в некоторой степени функционировали в отношении нитрогеназной активности в E. coli, когда другие белки Nif экспрессировались как полипептиды дикого типа, причем некоторые с повышенной активностью. Наблюдаемые вариации показали, что каждый полипептид Nif обладал внутренними характеристиками, которые влияли на количество накопленного полипептида, его транспортировку и процессинг с помощью MPP. Критические элементы NifH и NifK были легко экспрессированы и обнаружены, а для активности нитрогеназы, как известно, необходим высокий уровень этих белков. Однако они были нерастворимы в экспериментах с листьями, наряду с NifB, NifD, NifE, NifJ и NifV. В этих экспериментах, за исключением NifD, слитой полипептид NifY экспрессировался на самом низком уровне среди полипептидов Nif. Некоторые полипептиды Nif были успешно расщеплены MPP внутри матрицы и накапливались до более высоких уровней по сравнению с их цитоплазматическими аналогами, что позволяет предположить, что локализация в митохондриях была способом стабилизации слитых полипептидов после расщепления MPP. Слитой полипептид MTP::NifQ плохо расщеплялся, возможно потому, что белок-предшественник NifQ обладал меньше способностью проникать в митохондриальный матрикс из-за устойчивости к разворачиванию или неправильному нацеливанию.
В этих экспериментах слитой полипептид, имеющий NifH из K. oxytoca, был нерастворим в митохондриальном матриксе растения. Поскольку NifM может потребоваться для стабильности и растворимости NifH в бактериях (Lei et al., 1999; Howard et al., 1986), в следующем эксперименте была исследована комбинация митохондриально нацеленных NifH и NifM в анализе транзиентной экспрессии листьев.
Слитой полипептид NifB K. oxytoca был нерастворим при локализации в митохондриях, что согласуется с результатами, описанными для NifB A. vinelandii при нацеливании на митохондрии дрожжей и растения (Burén et al 2017a).
Рассматривая эти данные вместе, изобретатели пришли к выводу, что 7 слитых полипептидов Nif, а именно NifF, NifN, NifS, NifU, NifW, NifY и NifZ, экспрессировались с хорошими уровнями, процессировались эффективно и локализовались в митохондриальном матриксе в преимущественно растворимой форме, хотя содержание NifY было относительно низким. Эти N-концевые слитые полипептиды после расщепления MPP сохраняли допустимые уровни активности (Таблица 6).
Таблица 6. Влияние пептида «scar» (scar9) из девяти аминокислот pFAγ51, трансляционно слитого с отдельными белками Nif, на функцию нитрогеназы в E. coli. Значения представлены как % активности восстановления ацетилена по сравнению с pMIT2.1, pB-ori, негативной регуляцией, ΔNifM, кодирующей последовательностью NifM, удаленной из pMIT2.1.
(% pMIT2.1)
Пример 5. Обнаружение слитых полипептидов Scar9-Nif
Для обнаружения специфичных слитых полипептидов, экспрессированных в бактериальной системе, использовались способы жидкостной хроматомасс-спектрометрии (LC-MS). Этот способ сочетал в себе возможности физического разделения жидкостной хроматографии с возможностями масс-спектрометрического анализа (МС) для обнаружения специфичных пептидов, продуцированных при расщеплении белковых экстрактов трипсином.
Штамм JM109 E. coli, отдельно содержащий каждый из модифицированных векторов pMIT2.1 вместе с pN249, культивировали, а белки экстрагировали, как описано в Примере 1. Образцы белка хранили при -20°C перед восстановлением, алкилированием и триптическим расщеплением. Образцы белка восстанавливали, алкилировали и обрабатывали трипсином с использованием протокола подготовки образцов с фильтрацией (FASP), как описано в Примере 1, и анализировали способом LC-MS, как описано в Примере 1. Исследованные образцы перечислены в Таблице 7. Каждая генетическая конструкция для образцов 5-19 кодировала один модифицированный полипептид Nif, а другие 15 полипептидов Nif были дикого типа, как для K. oxytoca. Образцы 1-4 не содержали полипептидов, включая scar9.
Таблица 7. Генетическая конструкция, использованная для обнаружения слитого полипептида методом LC-MS
nd: не проводилось
Первоначально 4 образца оценивали на эффективность расщепления трипсином. Образцы 5 (NifB) и 6 (NifE) обрабатывали трипсином в течение двух инкубационных периодов, 30 мин и в течение ночи (16-18 ч). Из каждого образца, 4 мкл триптических пептидов вводили в масс-спектрометр 6600 Triple TOF с использованием жидкостного хроматографа Eksigent microLC (85 мин). Данные обрабатывались в ПО ProteinPilot на основе видоспецифичных баз данных UniProt Knowledgebase (UniProtKB), к которым добавлялись пользовательские базы данных и базы данных загрязнителей: Uniprot-Swiss Prot E. coli+Пользовательская база данных (Mit2Nif) + Общий репозиторий дополнительных белков; Mit2Nif+Mit2.1 Nif-Scar. Эти базы данных включали все предсказанные пептиды, продуцированные при расщеплении белков Nif трипсином. Ожидалось, что образцы белков 5 и 6 из конструкций, кодирующих FAγ-Scar9-NifB и FAγ-Scar9-NifE, будут содержать 16 белков Nif, 15 из которых являются белками дикого типа, а шестнадцатый, имеющий scar9 будет представлять собой NifB и NifE, соответственно.
Более короткая продолжительность расщепления трипсином, равная 30 мин, дала больше идентификаций белков/пептидов, чем более длительное расщепление. Полную панель образцов E. coli (№ 1-19) затем расщепляли трипсином в течение 1 часа, а не расщепляли в течение ночи.
Были исследованы идентификации пептидов для N-концевой последовательности scar9. Ограниченное свидетельство IDA (6600TF LC-MS/MS) полностью расщепленного пептида MSTQVVR (SEQ ID NO:55) и полутриптического пептида MSTQVVRNR (SEQ ID NO:49) получили при низкой достоверности идентификации пептида. Пептиды, имеющие либо немодифицированный, либо окисленный остаток метионина, также оценивали с использованием MRM. Однако эти пептиды не удалось подтвердить в исследуемых образцах ни с помощью поиска 6600TF LC-MS/MS и базы данных ПО ProteinPilot, ни с помощью целевого MRM 6500 QTRAP LC-MS/MS.
Было выдвинуто предположение, что метионин, инициирующий трансляцию, мог быть посттрансляционным отщеплением в бактериях, как объяснение низкой достоверности идентификации пептидов. Когда рекомбинантные белки экспрессируются в бактериальных системах экспрессии, считается, что инициирующий метионин часто отщепляется метионин-аминопептидазой (MAP) с эффективностью, зависящей от размера остатка, примыкающего к N-метионину (Hirel 1989, Xiao 2010). Когда остаток в позиции 2 был остатком Ser, как в случае для полипептидов FAγ-scar9-Nif, то было подсчитано, что N-концевой Met часто отщеплялся (эффективность 84%).
Таким образом, дополнительные модифицированные пептиды оценивались на основе слитых полипептидов Nif: STQVVR (+1, +2) (SEQ ID NO:50) и полутриптический пептид STQVVRNR (+2, +3) (SEQ ID NO:56). Пептид STQVVR (SEQ ID NO:50) был коротким и не был идентифицирован в предыдущих анализах, вероятно, по трем причинам. Во-первых, он имел массу (688 Да), которая давала бы значение m/z (345,2, +2), которое ниже, чем установлено в стандартных параметрах LC-MS (диапазон m/z 350-2000), во-вторых, он имел низкую гидрофобность и следовательно, он мог не сохраняться в колонке, и, в-третьих, он был слишком коротким, чтобы алгоритмы поиска в базе данных могли уверенно сопоставить последовательность. Первоначально, образцы № 1-19 (Таблица 7) были объединены и обработаны в 6600TF LC-MS/MS в различных условиях, напр., при снижении диапазона масс m/z от 350 до 300 и при расширении отслеживаемых состояний заряда для включения +1, а не только от +2 до +5, а также путем определения списков включений, которые инкапсулировали прогнозируемые целевые массы. Ни одно из этих изменений не привело к положительной идентификации STQVVR (SEQ ID NO:50) ни в спектральных данных, ни в поисках по базе данных.
Затем оценивали триптический пептид STQVVR (SEQ ID NO:50) и полутриптический пептид STQVVRNR (SEQ ID NO:56) с использованием селективной регистрации избранных реакций распада нескольких ионов (MRM) в 6500 QTRAP с использованием 4 транзиций с 2 состояниями заряда. Это давало пик для STQVVR (SEQ ID NO:50), который исследовали с помощью сканирования Enhanced Production Ion (EPI) для получения полных спектров сканирования МС/МС для целевого MRM. Это подтвердило присутствие модифицированного укороченного N-концевого пептида, лишенного N-концевого Met. Обнадеживает тот факт, что специфичные полипептиды FAγ-Scar9-Nif могут быть обнаружены этим методом в сложных смесях белков.
Затем этот метод использовали для сравнения уровня экспрессии каждого из различных полипептидов FAγ-Scar9-Nif при экспрессии из модифицированных векторов pMIT2.1 в E. coli. Для оценки образцов из JM109 был разработан комплексный метод MRM с 230 транзициями (Таблица 7). Сюда входили пептиды с высоким уровнем отклика (4 транзиции/пептид), идентифицированные для следующих белков Nif: B, D, E, F, H, K, M, N, Q, S, U, W, Y и Z. Также были включены контрольные пептиды из FAγ-Scar9 и белок хлорамфеникол-ацетилтрансферазы (CAT). Содержание пептида, специфичного к CAT, в каждом образце измеряли для стандартизации уровней Nif между различными конструкциями, как описано в Примере 1. Были приняты меры для использования равных количеств общего белка в каждом из образцов. Содержание обнаруженного CAT-специфичного пептида было одинаковым во всех экспериментальных образцах, что указывает на то, что содержание полипептидов Nif, образованных в разных образцах с помощью системы анализа pN249/pMIT2.1, можно сравнивать должным образом. Содержание пептида STQVVR (SEQ ID NO:50), полученного из FAγ-Scar9, было наибольшим в образцах 9 (FAγ-Scar9-NifH) и 11 (FAγ-Scar9-NifM), оба из которых показали сильную экспрессию относительно других, а за ними следуют образцы 10 (FAγ-Scar9-NifK), 14 (FAγ-Scar9-NifS) и 15 (FAγ-Scar9-NifU). Более низкое содержание было обнаружено в других образцах 5-19, за исключением, возможно, NifV. Пептид STQVVR (SEQ ID NO:50) отсутствовал в образцах 1-4 с негативной регуляцией, как ожидалось из-за отсутствия последовательности MTP-FAγ-scar9.
Содержание полипептидов scar9::NifD, scar9::NifK, scar9::NifH, scar9::NifS и scar9::NifM в клетках E. coli измеряли с помощью нацеленной масс-спектрометрии селективной регистрации избранных реакций распада нескольких ионов (MRM-MS), как описано в Примере 1. Измерения показали, что содержание специфичных пептидов для слитого полипептида NifS было примерно одинаковым во всех образцах. Напротив, наибольшее отклонение было обнаружено для scar9::NifM, где содержание слитого полипептида NifM было примерно в 50 раз увеличено по сравнению с образцами, в которых экспрессировался NifM дикого типа. Аналогичным образом, но в меньшей степени, пептид scar9, слитой с NifH, приводил к 2-3-кратному увеличению содержания NifH по сравнению с содержанием NifH дикого типа в других штаммах. В контрольных образцах, в которых был удален ген NifM (ΔNifM), пептиды, специфичные к NifM, как и ожидалось, не были обнаружены. Аналогичным образом, пептиды, специфичные для NifD, NifK и NifH, не были обнаружены в образцах из E.coli, содержащих pB-ori, где эти гены не присутствовали. Данные анализы также показали, что содержание NifD и NifK было достаточно стабильным во всех образцах, за тем заметным исключением, что при наличии scar9::NifY содержание NifD и NifK было снижено примерно до 30% от уровней, обнаруженных в штаммах, имеющие NifY дикого типа. Это снижение уровней NifD и NifK было подтверждено анализом вестерн-блот экстрактов из клеток E. coli с использованием антител, которые связывались с полипептидами NifD или NifK дикого типа. Авторы изобретения пришли к выводу, что добавление мотива scar9 к N-концу полипептидов Nif, представляющих продукт опосредованного MPP-расщепления слитых MTP-FAγ51 при экспрессии в E. coli, может влиять на уровень накопления полипептидов, сохраняя при этом хотя бы некоторую активность функции нитрогеназы.
В этих анализах содержание NifH-специфичных пептидов увеличивалось примерно в 2-3 раза по сравнению с контрольными клетками, когда и только когда в клетках продуцировался scar9-NifH. Напротив, NifS и NifE являлись примерами полипептидов, которые стабильно накапливались во всех векторах, полученных из pMIT2.1, где уровень 2 NifS-специфичных пептидов или 2 NifE-специфичных пептидов и пептида продолжения scar9, слитого с CAT, варьировался примерно в пределах 20% по всем образцам. Эти результаты показали, что N-концевые изменения полипептидов NifH и NifM значительно увеличивают содержание этих двух белков по сравнению со всеми другими белками Nif и CAT.
Эти результаты, а также результаты, приведенные в Таблице 4, дали некоторое представление об эффективности, измеренной с помощью ARA, продолжения scar9 в NifH, NifM и NifE относительно функции нитрогеназы. Хотя содержание полипептида scar9-NifH увеличивалось примерно в 2-3 раза у бактерий, содержащих pSO012, однако scar9-NifH показал 110% активность в анализе ARA по сравнению с диким типом. С другой стороны, scar9-NifM накапливал гораздо больше, по сравнению с диким типом, но анализ ARA показывал только примерно 10% активность по сравнению с контрольным образцом. Этот результат предполагает, что эти высокие уровни полипептида scar9-NifM могли действовать как негативный регулятор функции ARA.
Способ LC-MS использовали для обнаружения специфичных слитых полипептидов в растительных клетках (Пример 12), что показывает его общую применимость.
Пример 6. Экспрессия MTP-NifD K. oxytoca в клетках растений и дрожжей приводит к продуцированию вторичного продукта расщепления.
Предыдущее сообщение изобретателей показало, что из всех 16 полипептидов Nif наиболее сложным для продуцирования в растительных клетках являлся NifD (Allen et al., 2017). Они также сообщили, что, когда слитой полипептид MTP-FAγ::NifD::HA, имеющий аминокислотную последовательность NifD K. oxytoca дикого типа, продуцировался в клетках N. benthamiana, на мембранах вестерн-блоттинга проявлялись дополнительные полосы с более низкой молекулярной массой. Дополнительные полосы включали интенсивную полосу, массой ~ 48 кДа. Было высказано предположение, что эти дополнительные полосы соответствуют продуктам деградации слитого полипептида NifD, результату вторичного расщепления в скрытом сайте протеазы или, возможно, продукту альтернативных сигналов инициации транскрипции или трансляции.
Влияние изменения промоторов и последовательностей MTP
Чтобы подтвердить эти наблюдения и исследовать, связаны ли дополнительные полосы с комбинацией определенных промоторов или последовательностей MTP с последовательностью NifD, в конструкцию SN10 был внесен ряд генетических модификаций. Исходная конструкция SN10 кодировала слитой полипептид MTP-FAγ51::NifD::HA (SEQ ID NO:122), где аминокислотная последовательность NifD была такой, как указано в SEQ ID NO:18, и экспрессировалась с усиленным промотором e35S и с использованием кодон-оптимизации для N. benthamiana. В некоторых модификациях промотор e35S из SN10 был заменен другим промотором, например промоторами S4, S4v2 или S7 вируса карликовости подземного клевера (SCSV). В других случаях MTP-FAγ51 заменяли другим MTP, например MTP-L29 (SEQ ID NO:34) или MTP-CPN60 (SEQ ID NO:28). Конструкции, использованные в этом эксперименте, перечислены в Таблице 8 и включают некоторые из конструкций, описанных в Примере 2. Эти конструкции создавались методом сборки Golden Gate (Weber et al., 2011) со специфичными компонентами, как описано в работе Engler (2014). Некоторые из химерных генов схематично показаны на Рисунке 4 (верхняя панель).
Эти конструкции в A. tumefaciens инфильтрировали в клетки листьев N. benthamiana, как описано в Примере 1, и белковые экстракты анализировали вестерн-блоттингом с использованием антитела НА. Для каждой из конструкций парные инфильтрирования проводили либо без конструкции pRA25, либо при наличии pRA25 (кодирующей слитой полипептид MTP-FAγ::NifK; SEQ ID NO:57), поскольку совместная экспрессия NifK без С-концевого продолжения, как было показано, увеличивает содержание NifD (WO2018/141030). Репрезентативные анализы вестерн-блот представлены на Рисунках 4 и 5. Было замечено, что как MPP-процессированная, так и непроцессированная формы слитого полипептида были продуцированы для каждой конструкции, как и полипептид ~ 48 кДа. В каждом случае, когда присутствовал pRA25 (Рисунок 4, нижняя панель), интенсивность полосы ~ 48 кДа была больше, чем интенсивность процессированного полипептида MTP::NifD (полоса 2). Это также наблюдалось для всех вариантов с использованием другой последовательности MTP, полоса полипептида 48 кДа была наиболее интенсивной из полипептидных полос при анализе вестерн-блот, независимо от используемой последовательности МТР (Рисунок 5). Вновь было отмечено, что присутствие экспрессирующей конструкции MTP-NifK часто увеличивало содержание всех полипептидов NifD, включая доминантную полосу приблизительно при 48 кДа.
Была создана другая конструкция SN46, кодирующая слитой полипептид NifK. Эта конструкция имела усиленный промотор e35S и 5’-UTR, включая омега-фрагмент TMV для максимальной эффективности трансляции, последовательность 35S полиаденилирования/терминации транскрипции и кодирующий полипептид MTP-Su9::NifK, имеющий С-конец дикого типа (SEQ ID NO:58). В кодирующей области использовалась кодон-оптимизация для N. benthamiana, а не кодон-оптимизация в pRA25 для человека. Конструкцию SN46 сравнивали с pRA25 на предмет эффективности увеличения накопления слитого полипептида NifD после совместного инфильтрирования с конструкцией NifD. Было отмечено, что конструкция SN46 была, по меньшей мере, так же эффективна, как и pRA25, в увеличении накопления слитого полипептида NifD, но также приводила к накоплению продукта полипептида массой ~ 48 кДа. Репрезентативные анализы вестерн-блот представлены на Рисунке 6.
Поскольку полипептид массой ~ 48 кДа был обнаружен с использованием антитела HA, то он соответствовал C-концевому продукту расщепления протеазой транслированного гибридного полипептида. Эти результаты показали, что C-концевой полипептид массой ~ 48 кДа продуцировался в растительных клетках из слитого полипептида NifD K. oxytoca дикого типа независимо от промотора или последовательности MTP, использованных для его экспрессии. Полипептид массой ~ 48 кДа упоминается здесь как «продукт вторичного расщепления NifD» или как «продукт деградации» NifD.
Таблица 8. Генетические конструкции для исследования продуцирования и процессинга слитых полипептидов MTP-NifD в растительных клетках. Каждая конструкция кодировала слитой полипептид, включающий перечисленный MTP, слитой с N-концом последовательности NifD K. oxytoca дикого типа.
Вторичное расщепление связано с митохондриальным нацеливанием?
Изобретатели стремились определить причину вторичного расщепления/деградации NifD, во-первых, происходило ли оно до или после импорта митохондрий. Для исследования была создана конструкция (SN34) NifD, которая была идентична SN10, за исключением того, что последовательность MTP-FAγ51 была заменена меткой эпитопа HA, таким образом кодирующей слитой полипептид HA::NifD::HA. Этот полипептид, лишенный MTP, не будет нацелен на митохондрии, но вместо этого ожидается, что он будет локализован в цитоплазме растительных клеток. При наличии эпитопа HA на обоих концах продукта трансляции ожидалось, что любое внутреннее расщепление протеазой приведет к образованию N-концевого продукта и C-концевого продукта, которые оба могут быть обнаружены с помощью антитела HA, если они не будут подвергаться дальнейшей деградации. Была создана вторая генетическая конструкция, в которой С-концевая метка НА была удалена из SN34. Эта конструкция (SN33) кодировала слитой полипептид HA:NifD, который был почти идентичен по размеру MPP-процессированному полипептиду MTP-FAγ51::NifD, в которой каждый процессирует только одну метку эпитопа HA, что делает сравнение более прямым.
После совместного инфильтрирования SN75 и SN46 в N. benthamiana и анализа вестерн-блот белковых экстрактов из инфильтрированных клеток листа было обнаружено, что SN33 и SN34 продуцировали дискретные интенсивные полосы, соответствующие по размеру полноразмерным слитым полипептидам, транслированным из этих конструкций. Основная полоса полипептида для SN34 была немного больше, чем полоса полипептида для SN33, что, как предполагается, связано с присутствием дополнительного С-концевого эпитопа НА в SN34. Эти NifD-специфичные полосы конструкций SN33 и SN34 были значительно сильнее по интенсивности, чем соответствующая полноразмерная полоса, продуцированная из клеток, инфильтрированных с помощью конструкции SN10. Важно отметить, что после введения SN34 и SN33 не наблюдалось C-концевого продукта расщепления/деградации массой 48 кДа. Аналогично, для SN34 не наблюдалось N-концевого продукта расщепления.
Была создана дополнительная конструкция, обозначенная как SN66, которая имела мутирующую последовательность МТР, чтобы исследовать, требуется ли для продуцирования полипептида массой 48 кДа первое расщепление последовательности МТР с помощью МРР. Для этого MTP-FAγ51, кодированная в SN10, была модифицирован последовательностью идентичной длины, которая содержала область из 5 последовательных замен аланина в MTP и вторую область из 8 замен, которые делали бы ее устойчивой к процессингу митохондрий с помощью MPP. Специфичные замены приведены на Рисунке 7. Вторая область сканирования аланином охватывала сайт распознавания и расщепления для MPP, и поэтому предсказывалось, что процессинг MPP будет отменен из-за этих замен. Неизвестно, будет ли этот слитой полипептид транспортироваться в митохондрии. Когда эту конструкцию вводили в клетки листа N. benthamiana, то по данным анализа вестерн-блот, белковые экстракты из клеток содержали продукт массой 48 кДа.
Была создана вторая конструкция, обозначенная SN64, имеющая аналогичную мутирующую последовательность MTP, имеющую замены аланина, по сравнению с последовательностью MTP-CPN60 (SEQ ID NO:28). Когда эту конструкцию исследовали на клетках листа N. benthamiana, снова наблюдали продукт вторичного расщепления массой 48 кДа (Рисунок 6).
Вместе эти результаты продемонстрировали, что вторичное расщепление/деградация слитых полипептидов MTP::NifD было следствием митохондриального нацеливания и, как предполагалось, вызвано митохондриальной протеазой. Однако вторичное расщепление не зависело от предшествующего расщепления последовательности MTP с помощью MPP в митохондриях.
Обнаружение N-концевого продукта расщепления NifD продемонстрировало, что вторичное расщепление происходит в специфичном сайте с помощью эндопротеазы.
Поскольку продукт C-концевого расщепления/деградации массой 48 кДа явно продуцировался в растительных клетках после введения SN10 и других конструкций, кодирующих слитые полипептиды MTP::NifD, то изобретатели хотели посмотреть, может ли быть соответствующий N-концевой продукт расщепления NifD наблюдаться в растительных клетках или происходит ли деградация под действием экзо-протеазной активности с N-конца. Соответственно, была создана другая конструкция (SN75), которая была идентична SN10, за исключением того, что Gly-Gly-связанная метка HA также была включена непосредственно после MTP-FAγ51 и перед кодирующей областью NifD, а SEQ ID NO:36 использовали в качестве MTP-FAγ51. Было предсказано, что если слитой полипептид, полученный из этой конструкции, будет расщеплен в одной и той же специфичной локализации в пределах NifD, то будут продуцированы два продукта с меткой HA - более длинный C-концевой продукт массой ~ 48 кДа, ранее наблюдавшийся в экстрактах MTP::NifD, и более короткий N-концевой продукт массой ~ 13кДа. Однако, учитывая, что специфическая пептидаза в митохондриях разлагает N-концевые препоследовательности после расщепления MPP (Kmiec et al, 2013), то изобретатели не знали, будет ли наблюдаться какой-либо продукт N-концевого расщепления/деградации.
После инфильтрирования SN75 в листья N. benthamiana и анализа вестерн-блот белковых экстрактов был обнаружен более короткий N-концевой продукт массой примерно 15 кДа, а также более длинный C-концевой продукт массой примерно 48 кДа. Хотя суммарные размеры этих двух продуктов и были немного больше, чем прогнозируемый размер MPP-процессированного полипептида MTP-FAγ51::HA::NifD::HA (57,6 кДа), то это отклонение, вероятно, было результатом завышенной оценки размеров полос относительно маркеров, которые могли быть связаны с поверхностными зарядами полипептидов, влияющими на скорость миграции при электрофорезе на геле. Тем не менее, этот результат продемонстрировал, что вторичное расщепление части слитого полипептида NifD было специфичным и дискретным, происходящим в специфичном сайте полипептида NifD, а не результатом последовательной деградации с N-конца.
Происходит ли вторичное расщепление/деградация митохондриально-нацеленного NifD в дрожжах?
В работе Burén et al. (2017b) указано, что нацеливание полипептида NifD Azotobacter vinelandii на митохондрии дрожжей продуцирует более быструю миграцию полосы массой ~ 50 кДа, обнаруживаемой антителами к NifD. Авторы настоящего изобретения хотели определить, проявляют ли оптимизированные для растений последовательности NifD K. oxytoca аналогичное расщепление при экспрессии в дрожжах. Для этой цели был создан вектор экспрессии дрожжей, который включал кодирующую последовательность MTP-FAγ51::NifD::HA из SN10 с фланкирующими сайтами рестрикции KpnI/SacI, чтобы сделать возможным клонирование в вектор экспрессии дрожжей pYES2. Данную конструкцию обозначили SNY10. В качестве контроля за немитохондриальной локализацией была создана вторая дрожжевая конструкция NifD, обозначенная SNY196, в которой MTP-FAγ51 из SNY10 был заменен меткой эпитопа 6xHis. Эта вторая конструкция была разработана для экспрессии локализованного в цитоплазме полипептида NifD почти того же размера, что и процессированные полипептиды из SN10 или SNY10, что позволяет визуализировать ожидаемый размер с помощью анализа вестерн-блот. Также была создана растительный ортологичная конструкция SNY196 (SN196), в которой промотор GAL1 был заменен промотором e35S. Эта конструкция была идентична SN10, за исключением метки 6xHis, замененной на MTP-FAγ51 из SN10.
Дрожжевые клетки, содержащие либо конструкцию SNY10 (MTP-FAγ51::NifD: HA), либо конструкцию SNY196 (6xHis::NifD::HA), выращивали, как описано в Примере 1, для экспрессии генов, кодирующих слитые полипептиды. Белки экстрагировали из трансформированных клеток после индукции экспрессии трансгена и проводили анализ вестерн-блот с антителом HA. Результаты приведены на Рисунке 8. Для MPP-процессированного полипептида MTP-FAγ51::NifD::HA в дорожке для SNY10 наблюдалась полоса ожидаемого размера (~ 58 кДа) с менее интенсивной окраской. Этот полипептид имел тот же размер, что и растение, экспрессировавшее полипептид MTP-FAγ51::NifD::HA после процессинга MPP, и полипептид из SN196. Важно отметить, что гораздо более интенсивная полоса полипептида массой ~ 48 кДа наблюдалась для SNY10, которая была того же размера, что и у растения, экспрессировавшего продукт расщепления/деградации из SN10. То есть большая часть дрожжей, экспрессировавших MTP-FAγ51::NifD::HA, расщеплялась аналогично расщеплению в растительных клетках, в действительности даже более эффективно в клетках дрожжей. Тот факт, что продукты C-концевого расщепления дрожжевых и растительных клеток были одинакового размера, указывало на то, что расщепление протеазой происходило в одном и том же сайте митохондрий как дрожжевых, так и растительных клеток. Напротив, белковый экстракт из дрожжевых клеток, содержащий SNY196, продуцировал единственную дискретную полосу ожидаемого размера для немитохондриально нацеленного NifD. Не было обнаружено специфичной C-концевой полипептидной полосы NifD::HA из SNY196 с массой ~ 48 кДа, что указывало бы на немитохондриальное расщепление в том же сайте.
Примечательно, что в белковых экстрактах дрожжевых клеток, содержащих SNY10, не было обнаружено слитого полипептида MTP::NifD, который не подвергался процессингу с помощью MPP, в отличие от наблюдения, что в клетках N. benthamiana, продуцирующих тот же полипептид MTP-FAγ51::NifD, наблюдались как непроцессированные и MPP-процессированные формы полипептида. То есть, последовательность MTP в дрожжах была полностью процессирована MPP. Считалось, что это происходит из-за различий в механизме процессинга и эффективности среди двух организмов. Это могло также иметь место из-за того, что дрожжевые клетки были стабильно трансформированными клетками в отличие от растительных клеток, которые трансформировались только транзиентно.
Вместе эти результаты показали, что полипептид NifD дикого типа из K. oxytoca, экспрессированный как полипептид слитой с MTP, расщеплялся в одном и том же специфичном сайте при нацеливании на митохондрии дрожжей или растения и что расщепление зависело от митохондриального нацеливания.
Пример 7. Идентификация вторичного сайта расщепления в NifD дикого типа
Результаты экспериментов, описанных в Примере 6, показали, что вторичное расщепление слитого полипептида MTP::NifD происходило в специфичном сайте в последовательности NifD дикого типа и является следствием митохондриального нацеливания. Поскольку расщепление считалось нежелательным по нескольким причинам, то изобретатели хотели модифицировать область NifD в попытке предотвратить расщепление в растительных клетках. Исходя из размеров N-концевых и С-концевых продуктов расщепления, предполагалось, что сайт расщепления локализуется в области аминокислот 80-120 последовательности NifD дикого типа (SEQ ID NO:18). Однако существует возможность того, что на расщепление в специфичном сайте влияют дистальные последовательности, а не только аминокислоты, прилегающие к сайту расщепления. По этой причине изобретатели применили более широкий подход к идентификации специфичного сайта вторичного расщепления и окружающих аминокислот и возможных дополнительных областей, которые могут влиять на расщепление.
В качестве начальной попытки идентифицировать сайт расщепления в NifD или, по меньшей мере, предсказать его локализацию, в программное обеспечение Mitofate (Fukusawa et al., 2015) были введены как непроцессированные, так и MPP-процессированные аминокислотные последовательности, чтобы увидеть наличие какие-либо предсказанных сайтов MPP. Программное обеспечение Mitofate предсказывает сайты для расщепления с помощью MPP путем включения характеристик аминокислотной последовательности, включая положительно заряженные амфифильные соединения и мотивы препоследовательности, а также аминокислотный состав и физико-химические свойства. Программное обеспечение также предсказывает сайты расщепления препоследовательности с помощью MPP, путем создания матрицы весовых коэффициентов согласованной позиции между аминокислотными остатками -4 и +5 совмещенными сайтами расщепления из набора данных для формирования роста дрожжей. Этот инструмент также включает информацию о расстоянии от N-конца, так как MTP обычно имеют в длину от 10 до 90аа, а меньшую часть длиннее 110aa аминокислот (Huang et al., 2009).
Предполагая, что MPP может распознавать сайт вторичного расщепления после начального расщепления внутри MTP, поскольку препротеин, перемещенный через внешнюю и внутреннюю митохондриальные мембраны, и аминокислотные последовательности, полученные в результате начального события процессинга MPP, вводились в программное обеспечение Mitofates для двух длин MTP-FAγ, а именно FAγ-scar37-NifD (35aa FAγ scar плюс GG) и FAγ-scar11-nifD (9aa FAγ51 scar плюс GG). Анализ Mitofates с использованием последовательности FAγ-scar37-NifD дал предсказанный сайт расщепления сразу после аминокислоты G62 в последовательности VRGCAY (SEQ ID NO:60) относительно N-конца NifD, а с использованием последовательности FAγ-scar11-NifD дал предсказанный сайт расщепления сразу после N99 в последовательности RAGRRNYYTG (SEQ ID NO:61). Таким образом, анализ Mitofate показал, что последовательность NifD в этой области, по-видимому, обладает характеристиками одного или даже двух сайтов процессинга MPP. Как описано ниже, второй из этих предсказанных сайтов оказался пригодным для вторичного расщепления.
В другом подходе к идентификации областей в NifD, которые участвовали во вторичном расщеплении, был создан ряд генетических конструкций, каждая из которых имела блок из 5 последовательных аминокислотных замен в пределах приблизительной области вторичного расщепления NifD, где неаланиновые аминокислоты были заменены на аланины, а нативные аланиновые аминокислоты были заменены на глицины. То есть для всех замен использовали аланин, за исключением того, что остатки природного аланина заменяли глицином. Серия мутантных замещений охватывала около 6 кДа предполагаемого сайта расщепления от аминокислоты 49 до аминокислоты 108 SEQ ID NO:18. Эти конструкции были обозначены NifD-Var от 1 до 6 и Var от 9 до 14 (Таблица 9). Два других варианта имели дискретные замены на основе прогноза ПО Mitofates возможного сайта расщепления в последовательности VRGCAY (SEQ ID NO:60), обозначенного NifD-Var 7 и Var 8. Во всех других отношениях эти конструкции, кодирующие варианты NifD, были идентичны SN10 в том, что полипептиды имели MTP-FAγ51, трансляционно слитую с кодирующей белок NifD областью, и С-концевую метку эпитопа НА, которая позволяла обнаруживать любой продукт расщепления С-конца NifD.
Эти 14 конструкций были введены из A. tumefaciens индивидуально в клетки листа N. benthamiana вместе с SN46 (MTP-Su9::NifK). Белковые экстракты получали из инфильтрированной пятнистости листьев и подвергали электрофорезу SDS-PAGE и анализу вестерн-блот с использованием антитела HA. Из 14 исследованных вариантов 12 по-прежнему продуцировали продукт расщепления массой 48 кДа и были неотличимы по характеру образования их полос от полос, полученных из конструкции SN10, имеющей последовательность NifD дикого типа. Однако NifD-Var 13 (генетическая конструкция SN100) заметно не показывала продукта расщепления массой 48 кДа и, судя по размеру и интенсивности полосы при анализе вестерн-блот, имела относительно более высокое соотношение процессированного и непроцессированного FAγ51::NifD, чем другие варианты. Для NifD-Var 12 (SN99) была обнаружена слабая полоса массой 48 кДа, значительно меньшая по интенсивности, чем у дикого типа. Опять же, соотношение NifD, процессированного MPP, к непроцессированному NifD было больше для NifD-Var 12 по сравнению с диким типом и вариантами, отличными от NifD-Var13. На основании аминокислот, замененных в NifD-Var 12 и 13, был сделан вывод, что специфичная область полипептида NifD, включающая, по меньшей мере, некоторые аминокислоты в аминокислотной последовательности RAGRRNYYTG (SEQ ID NO:61), соответствующие аминокислотам 94-103 из SEQ ID NO:18, требуется для вторичного расщепления NifD в митохондриях.
На основании этого эксперимента и сделанного вывода были созданы генетические конструкции, кодирующие второй набор аминокислотных вариантов NifD, в которых были заменены одна, две или три аминокислоты в последовательности RAGRRNYYTG (SEQ ID NO:61). В этом наборе вариантов, вместо аминокислот дикого типа не использовались аланины, а скорее были внесены изменения, основанные на филогенетическом анализе большого набора встречающихся в природе последовательностей NifD (см. ниже), а моделирование структур NifD-NifK использовалось для идентификации замены аминокислот в каждой конкретной позиции. Идея заключалась в том, что природные варианты последовательности RAGRRNYYTG (SEQ ID NO:61) с большей вероятностью могут поддерживать функцию NifD и что рациональная конструкция вариаций возможна, чтобы избежать вторичного расщепления и поддерживать функцию. Каждая конструкция была идентична SN10, за исключением аминокислотной замены (замен), поэтому кодирование полипептида, имеющего MTP-FAγ51 слитой с NifD и С-концевую метку эпитопа НА, позволяло обнаруживать продукт расщепления С-конца массой 48 кДа. Замены в этом наборе вариантов NifD, обозначенных NifD-Var от 15 до 36, перечислены в Таблице 10, а репрезентативный анализ вестерн-блот показан на Рисунке 9.
19 индивидуальных генетических конструкций (SN108-SN126), каждая из которых кодирует один из вариантов последовательностей NifD, вводили в клетки N. benthamiana с помощью A. tumefaciens, а через 5 дней белки экстрагировали для экспрессии химерных генов и подвергали электрофорезу SDS-PAGE и анализу вестерн-блот с использованием антитела HA. Как было сделано ранее, генетическая конструкция SN46, кодирующая MTP-Su9::NifK, была совместно инфильтрирована с каждым вариантом NifD для увеличения уровня накопления NifD. Исходя из анализа вестерн-блот наблюдали три группы вариантов: (1) Группы, которые показали идентичный характер исчерченности, полученный с SN10, содержащей последовательность NifD дикого типа, а именно SN108, SN109, SN111-113, SN115, SN116 и SN121. Для них соотношение интенсивностей полосы массой 48 кДа к MPP-процессированному NifD (первичное расщепление) было, по существу, таким же, как и для SN10, что указывает на то, что аминокислотная замена (замены) не влияли на вторичное расщепление. (2) Группы, которые показали продукт массой 48 кДа, но соотношение интенсивностей продукта массой 48 кДа к NifD, процессированному MPP, было заметно снижено по сравнению с соотношением для SN10 (SN110, SN122 и SN123). (3) Варианты, которые показали отсутствие вторичного продукта расщепления/деградации массой 48 кДа (SN114, SN117, SN118, SN119, SN120, SN124, SN125 и SN126), то есть вторичное расщепление было исключено или уменьшено до такой степени, что не обнаруживалось путем 1-3 специфичных аминокислотных замен. Наиболее примечательно то, что два варианта из этого последнего набора, а именно NifD-Var 21 с заменой Y100Q (кодировался SN114) и NifD-Var29 с заменой Y100K (SN119), имели одиночные аминокислотные замены, а другой вариант Var 24, кодированный SN117, имел две аминокислотные замены YY100-101QT. То, что эти специфичные аминокислотные замены будут иметь такой эффект, предсказать заранее было невозможно.
Набора вариантов показал, что замена аргинина в позиции 98 сама по себе не предотвращает вторичного расщепления (NifD-Var 19 и Var 32). Аналогично, одиночные аминокислотные замены аспарагина в позиции 99 (NifD-Var20), тирозина в позиции 101 (NifD-Var 15 и Var 22), треонина в позиции 102 (NifD-Var 16 и Var 23) или 2 или 3 замены в позициях 101-103 не препятствовали вторичному расщеплению. Однако, исследованные одиночные, двойные или тройные замены, включая тирозин в позиции 100 (NifD-Var 21, 24, 26, 29 и 30), все устраняли вторичное расщепление NifD. Расщепление также устранялось двойными или тройными заменами аминокислот, не включая тирозин в позиции 100 (NifD-Var 34, 35 и 36). Было ясно, что несколько вариантов могут быть легко идентифицируемы при наличии аминокислотных замен в позициях, выбранных из аминокислотных позиций 98-102, которые были устойчивы к вторичному расщеплению, например, с использованием подхода, приведенного здесь в качестве примера.
Устранение вторичного расщепления MTP::NifD в дрожжах
Учитывая данные из Примера 6 о том, что расщепление слитого полипептида MTP::NifD происходило в той же области в дрожжевых клетках, что и в митохондриях растений, вариант с заменой Y100Q был исследован в митохондриях дрожжей. С этой целью кодирующая белок область из SN114 (MTP-FAγ51::NifD (Y100Q)::HA) была амплифицирована с помощью PCR для создания фланкирующих сайтов рестрикционных ферментов KpnI и SacI, которые были использованы для вставки гена в вектор экспрессии дрожжей pYES2. Эта конструкция для экспрессии дрожжей была обозначена как SNY114. Белковые экстракты получали из трансформантов дрожжей, содержащих SNY114, и подвергали анализу вестерн-блот. Примечательно, что экстракты из клеток, содержащих SNY114, давали интенсивную полосу того же размера, что и у конструкции NifD-Var 29 в растительных клетках, но с гораздо меньшим содержанием вторичного расщепления. Это сильно контрастировало с результатом на Рисунке 8 из последовательности NifD дикого типа, которая в ходе экспрессии в дрожжах продуцировала интенсивный продукт расщепления/деградации массой 48 кДа. Хотя наблюдались некоторые полосы белка других размеров, наблюдаемые у SNY114, однако они были менее интенсивными, чем преобладающая полноразмерная полоса, соответствующая требуемому MPP-процессированному полипептиду MTP::NifD::HA. Был сделан вывод, что полноразмерный корректно процессированный полипептид NifD экспрессировался как преобладающий полипептид MTP::NifD как в митохондриях дрожжей, так и в митохондриях растений, когда аминокислотные замены производились в позициях 98-102 NifD со ссылкой на SEQ ID NO:18, например, в позиции 100.
Таблица 9. Варианты замены на аланин в слитом полипептиде FAγ51-NifD и их влияние на вторичное расщепление/деградацию в растительных клетках
Таблица 10. Второй набор вариантов аминокислотной замены слитого полипептида FAγ51-NifD и влияние на вторичное расщепление/деградацию и влияние на функцию в бактериальных анализах ARA. Последовательность дикого типа RAGRRNYYTG (SEQ ID NO:61) была заменена указанной модифицированной последовательностью. Nt= не исследовалось.
Демонстрация сайта вторичного расщепления способом масс-спектрометрии
Белковые экстракты из листьев N. benthamiana, инфильтрированные SN14 (MTP-Su9::NifD::HA), подвергали электрофорезу SDS-PAGE с использованием геля, имеющего концентрацию полиакриламида 4-20% (Invitrogen). Гель окрашивали синим красителем (Bulldog Bio). После обесцвечивания в воде, из геля отрезали 5 срезов в области с молекулярной массой 37-50 кДа. Срезы пронумеровывали от 1 до 5 от меньшего молекулярного веса к большему. Каждый срез геля разрезали на кубики размером примерно 1 мм и вымачивали в 150 мкл 30% метанола в течении 15 минут. Для уменьшения количества окисленных белков, буфер удаляли и заменяли на 100 мкл буфера со свежими 25 ммоль бикарбоната аммония (ABC) и с 5 мкл 15% дитиотреитола и инкубировали при комнатной температуре в течение часа. Остатки цистеина инактивировали добавлением 5 мкл 40% акриламида и инкубировали при комнатной температуре в течение 1 часа, после чего буферы осторожно удаляли. Выполнялись три стадии промывки, каждая из 50 мкл буфера ABC и 50 мкл ацетонитрила с инкубацией при комнатной температуре. Срезы геля сушили с добавлением 100 мкл 100% ацетонитрила в течение 2 минут, который после утилизировали. Затем белки в срезах высушенного геля расщепляли 0,1 мкг трипсина (Promega) в 20 мкл ABC с инкубацией в течение ночи при 37°C. Расщепление трипсином останавливали 1 мкл 50% (об./об.) раствора муравьиной кислоты и обрабатывали ультразвуком в течение 15 мин. Образцы фильтровали после добавления 10 мкл воды перед переносом во флаконы для LCMS.
Полученный триптический гидролизат из каждого среза геля вводили в систему Dionex Nanomate 3000 (ThermoFisher) высокоэффективной жидкостной хроматографии (LC), непосредственно связанную спектрометром Orbitrap Fusion Tribrid Mass. Пептиды обессоливали в течение 5 минут в предколонке Acclaim PepMap C18 (300 Å, 5 мм x 300 мкм) при скорости потока 10 мкл/мин с загрузочным растворителем и разделяли в Acclaim PepMap C18 (100 Å, 150 мм × 0,075 мм) со скоростью потока 0,3 мкл/мин при 35 °C. Использовали линейный 5%-40% градиент растворителя B в течение 60 мин с последующей промывкой и повторным уравновешиванием при 40-99% B в течение 5 мин, выдержкой 5 мин при 99% B, с возвратом к 5% B в течение 6 мин и выдержкой 7 мин. Использовали следующие растворители: (А) 0,1% муравьиной кислоты и 99,9% воды; (B) 0,08% муравьиной кислоты, 80% ацетонитрила и 19,92% воды. Высокоэффективный LC был напрямую соединен с ионным источником Nanospray Flex Ion системы Orbitrap Fusion MS. Напряжение ионного распыления было установлено равным 2400 В, давление продувочного газа было установлено в 1 Arb, а температура трубки для переноса ионов была установлена на 300 °C. Данные были получены в режиме сбора данных, зависящем от данных, находящихся в обзорном сканировании Orbitap-MS с последующим параллельным получением сканирования Orbitrap с высоким разрешением 120 000 и множественных событий MS/MS в линейной ионной ловушке в течение 3-секундного периода. Анализ МС на первом этапе выполняли в режиме положительных ионов в диапазоне масс m/z от 400-до 1500 с мишенью AGC 4×105 и максимальным временем впрыска 50 мс. Тандемные масс-спектры получали в ионной ловушке на ионах-предшественниках, которые превышали порог интенсивности в 1 000 отсчетов с состоянием заряда 2-7. Спектры получали с использованием квадрупольной изоляции с изоляционным окном 1,6 m/z и HCD (Высокоэнергетичная столкновительная диссоциация), установленным на 28% в зависимости от размера и заряда иона-предшественника для оптимальной фрагментации пептида. Скорость сканирования ионной ловушки устанавливали быстрой с мишенью AGC 4×103 и максимальным временем впрыска 300 мс, а прибор настраивали на использование максимального распараллеливаемого времени для впрыска ионов в ловушку в течение 3-секундного окна, пока орбитальная ловушка собирала МС-спектры высокого разрешения. Было установлено динамическое исключение для исключения ионов-предшественников после одного появления с интервалом в 15 секунд и допуском по массе в 10 ч/млн.
Анализ данных для идентификации белков проводили с использованием алгоритма Sequest в Proteome Discoverer v2.2 (ThermoFisher). Карбамидометил использовали в качестве алкилирующего агента, а трипсин использовали в качестве фермента расщепления. Для окисления NifD использовали динамические модификации, максимум три модификации. Данные тандемной масс-спектрометрии анализировали по базе данных триптических пептидов для NifD, полученных из аминокислотной последовательности слитого полипептида, кодированной SN14 и протеомом N. benthamiana, и по базам данных о распространенных загрязнителях и специфичных организмах, аннотированных в UniProt. Результаты поиска по базе данных обрабатывали для идентификации белков с использованием 1% от средней доли ложных отклонений гипотезы (FDR), определенной встроенным инструментом FDR в программном обеспечении Proteome Discoverer.
Из пяти образцов, представленных на масс-спектрометрию, пептиды NifD не были идентифицированы в образце, полученном из гелевого среза для наивысшей молекулярной массы, а именно в образце 5. Напротив, пептиды NifD были идентифицированы в других образцах 1-4. Наибольшее покрытие было у образца 2, на второй самой нижней полосе, вырезанной из геля, с 17 специфичными триптическими пептидами, полученными из последовательности NifD, идентифицированной в этом образце. От 6 до 11 специфичных пептидов NifD были идентифицированы в образцах 1, 3 и 4. Важно отметить, что пептид YYTGVSGVDSFGTLNFTSDFQER (SEQ ID NO:100) был положительно идентифицирован в образце 2. Оценка XCorr была достаточно высокой для этого пептида, а оценка апостериорной вероятности ошибки (PEP) была достаточно низкой, чтобы подтвердить положительную идентификацию, что указывает на то, что ионы пептидного фрагмента не являлись продуктом пептида аналогичного размера, но являлись продуктом другого пептида. В ходе анализа был сделан вывод, что этот пептид должен происходить из SN14 путем специфичного расщепления последовательности NifD в последовательности RRNY (SEQ ID NO:101) между остатками аспарагина (N) и тирозина (Y) в растительных клетках, за которыми следует триптическое расщепление. Положительная идентификация сайта расщепления с помощью этого MS-анализа полностью согласуется с описанным выше мутационным подходом.
Пример 8. Филогенетический анализ NifD вокруг сайта вторичного расщепления
Ферменты нитрогеназы, включая полипептиды NifD, природным образом продуцируются во многих типах бактерий и архей. Набор из 1751 природной аминокислотной последовательности NifD, из очень широкого спектра бактериальных и архейных источников, был извлечен из базы данных InterPro 12 декабря 2018 года. Все последовательности были перечислены как члены семейства IPR005972, определенного как альфа-цепь молибден-железосодержащего белка нитрогеназы, которые все представляют собой полипептиды NifD типа молибден-железо. Последовательности принадлежали 21 разным типам. Большинство последовательностей были из Proteobacteria (63,0%), за которой следовали Firmicutes (12,3%) и Cyanobacteria (12,3%). Другие, но в меньших количествах, были из архей: Actinobacteria, Aquificae, Bacteroidetes, Candidatus Margulisbacteria, Candidatus Sumerlaeota, Chlorobi, Chloroflexi, Chordata, Chrysiogenetes, Deferribacteres, Elusimicrobia, Euryarchaeota, Fusobacteria, Lentisphaerae, Nitrospirae, Planctomycetes, Spirochaetes и Verrucomicrobia.
Набор из 1751 последовательности содержал 275 повторяющихся последовательностей. Повторяющиеся последовательности были удалены, в результате чего получился набор из 1476 уникальных последовательностей. Эти последовательности исследовали, чтобы понять разнообразие аминокислотных последовательностей в позициях, соответствующих последовательности RAGRRNYYTG (SEQ ID NO:61) K. oxytoca. Последовательности были выровнены с использованием программы множественного выравнивания последовательностей Mafft версии 7 с использованием стратегии FFT-NS-2 с параметрами по умолчанию, т. е. с использованием настроек по умолчанию «быстро и прогрессивно» (Katoh et al., 2013). Выровненные последовательности визуализировали с помощью программного обеспечения ALVIS (интерактивная неагрегативная визуализация и исследовательский анализ множественных выравниваний последовательностей) (Schwarz et al., 2016). Последовательности NifD имели в длину от 362 до 592 остатков. Множественное выравнивание последовательностей («мега-выравнивание») содержало 907 позиций, принимая во внимание многочисленные пробелы в индивидуальных последовательностях, которые были введены программой выравнивания. При мега-выравнивании предполагаемый сайт вторичного расщепления был обнаружен между позициями 270-275, соответствующими остаткам 97-102 в последовательности K. oxytoca (SEQ ID NO:18). Было идентифицировано шестьдесят-восемь последовательностей, которые содержали те же 10 аминокислотных последовательностей, что и аминокислоты 94-103 K. oxytoca (RAGRRNYYTG; SEQ ID NO:61).
Сеть сходства белков была создана для 1476 членов семейства InterPro IPR005972 с указанием кластеров родственных последовательностей из разных типов диазотрофов. Репрезентативные последовательности выбирались из различных кластеров (Таблица 11) и выравнивались в области, соответствующей аминокислотам 49-108 последовательности NifD K. oxytoca. Выравнивание для этой области показано на Рисунке 10. Была отмечена высокая степень консервативности последовательности, включая 19 аминокислот, которые были полностью консервативными, и многие другие, которые были высококонсервативными. На Рисунке 10 не показаны последовательности из Desulfotomaculum ferrireducens, Halanaerobium saccharolyticum, Clostridium ljungdahlii, Methanosarcina barkeri, Desulfovibrio vulgaris и Chlorobium tepidum, а родственные последовательности в их кластерах содержали вставку 50-60 остатков дальше по направлению к C-концу, с формированием подгруппы последовательностей NifD.
Частотное распределение остатков вычисляли вокруг предсказанного сайта вторичного расщепления, взятого сразу после аминокислот RRN, для позиций -3, -2, -1, +1, +2 и +3 (Таблица 12). Аргинин (R) в позиции -3 был полностью консервативным, за исключением двух последовательностей в наборе, которые показали «брешь» для обоих позиций -3 и -2. Однако эти две последовательности были только фрагментами, а не полными последовательностями NifD (A0A2N4YT47 - Klebsiella varicola, A0A2N5A8Y2 - Klebsiella varicola) и поэтому были неопределенными и могли быть исключены из дальнейшего анализа. Аргинин в позиции -2 был практически полностью консервативным. Было только две последовательности из 1476, которые содержали остаток, отличный от аргинина в этой позиции: NifD из Paenibacillus fujiensis (B9X2A1) содержал остаток цистеина, а NifD из Alcaligenes faecalis (Q44045) содержал остаток глицина. Неизвестно, были ли эти последовательности активными для NifD. Аспарагин (N) был высококонсервативным в позиции -1, присутствуя в 97,83% из 1476 последовательностей. Около 1,9% из 1476 последовательностей содержали остатки гистидина, фенилаланина, аланина или серина в этой позиции вместо аспарагина. Наиболее частым остатком в позиции +1 был тирозин (Y, 71,54%), за ним следуют глутамин, лейцин и лизин, каждый из этих трех с частотой, составляющей от 7 до 11%. Поскольку существовало значительное количество природных последовательностей NifD, содержащих одну из этих аминокислот, отличную от тирозина в этой позиции, то был сделан вывод о том, что эти аминокислоты в позиции +1 обеспечивают активность NifD. Фенилаланин, метионин и глутаминовая кислота также представлены в этой позиции с меньшими частотами. Наиболее частым остатком в позиции +2 был тирозин (64,43%), затем следовали аланин и треонин, а затем любая из шести других аминокислот с меньшими частотами. И снова был сделан вывод о том, что эти аминокислоты в позиции +2 обеспечивают активность NifD. Наиболее частым остатком в позиции +3 был валин (V, 27,24%), за ним следовали изолейцин, треонин и лизин, а затем любая из 11 других аминокислот. Очевидно, что степень консервативности аминокислот в шести позициях, соответствующих остаткам 97-102 последовательности NifD K. oxytoca, снизилась вдоль этой последовательности, от двух аргининов, которые считались важными для функции NifD, до позиции +3 и которые показали широкую изменчивость.
Аминокислотные последовательности NifD в количестве 1474 штук (за исключением двух частичных последовательностей, упомянутых выше) исследовали в позициях, соответствующих аминокислотам 97-101 SEQ ID NO:18, на наличие последовательности RRNY (SEQ ID NO:101), а более конкретно на наличие в этом наборе RRNYY (SEQ ID NO:102). В наличии было 1045 последовательностей (70,90%), которые содержали RRNY (SEQ ID NO:101), а из них 935 последовательности, содержали RRNYY (SEQ ID NO:102), соответствующую аминокислотам 97-101 из SEQ ID NO:18. На основе данных вторичного расщепления, описанных выше, был сделан вывод, что 1045 природных полипептидов NifD, имеющих последовательность RRNY (SEQ ID NO:101), будут подвергаться вторичному расщеплению в этой последовательности при вводе в митохондрии эукариотической клетки, тогда как полипептиды NifD, имеющие последовательность RRNX (SEQ ID NO:154), где X представляет собой любую аминокислоту, отличную от тирозина (Y), будут менее подвержены вторичному расщеплению в этой последовательности. Таким образом, эти последовательности NifD не являлись предпочтительными из-за их склонности к вторичному расщеплению. Напротив, последовательности NifD, содержащие любую аминокислоту, кроме тирозина (Y) в позиции, соответствующей Y100 NifD K. oxytoca (SEQ ID NO:18), являлись предпочтительными на основании того, что они, вероятно, будут устойчивы к расщеплению при введении в митохондрии эукариотической клетки. Такие последовательности можно легко исследовать для подтверждения их устойчивости к расщеплению в этой области при экспрессии в растительных клетках в виде слитого полипептида MTP-NifD.
При дальнейшем исследовании 1474 последовательностей было обнаружено 155 последовательностей (10,51%), имеющих последовательность RRNQ (SEQ ID NO:103), и 95 последовательностей (6,45%), имеющих последовательность RRNK (SEQ ID NO:104), обе из которых, как считалось, не подвержены вторичному расщеплению и поэтому более предпочтительны, чем последовательности, не содержащие глутамин или лизин в четвертой позиции. Эти полипептиды NifD считались более предпочтительными, чем полипептиды NifD, имеющие последовательность RRNF (SEQ ID NO:220). Затем было отмечено, что 141 из 155 последовательностей NifD, содержащих последовательность RRNQ (SEQ ID NO:103), содержала треонин (T) сразу после, то есть включала последовательность RRNQT (SEQ ID NO:105). Исходя из того, что полипептид, кодируемый Var 24 (SN117) и содержащий последовательность RRNQT (SEQ ID NO:105), не был расщеплен в этой позиции, и что последовательность относительно часто встречалась в природных полипептидах NifD, был сделан вывод, что полипептиды NifD, содержащие последовательность RRNQT (SEQ ID NO:105), были весьма предпочтительны для использования в митохондриях эукариотической клетки.
Таблица 11. Репрезентативные последовательности NifD из широкого спектра диазотрофных организмов
Таблица 12: Распределение частот аминокислотных остатков у 1476 природных последовательностях NifD вокруг вторичного сайта расщепления
* «X» означает неизвестную аминокислоту, присутствующую в последовательностях Methylocella palustris (Q6KCQ3) и Methylosinus trichosporium (Q6KCQ2).
Пример 9. Функциональные исследования вариантов NifD вокруг вторичного сайта расщепления
Варианты MTP-FAγ51::NifD, которые не показали расщепления в предполагаемом сайте между остатками 99 и 100, исследовали на функцию NifD в системе MIT2.1 в E. coli следующим образом. Чтобы ввести мутации, кодирующие аминокислотные замены, в ген NifD в pMIT2.1, и облегчить клонирование, сайты рестрикции для ферментов AgeI и SalI вводили в кодирующую область NifD, охватывающую сайты для аминокислотных изменений. Это выполняли с помощью PCR-опосредованного мутагенеза с использованием комбинаций олигонуклеотидных праймеров 5’-CTAATGCTACCGGTGAACGTAACCTGGCACTGATTCAAGAAGTACTGGAAGTGTTC-3’ (SEQ ID NO:108) и 5’-GTTACGTTCACCGGTAGCATTAGTCATCATCCGG CTCCTCCGCTAGATAAAAATGTG-3’ (SEQ ID NO:109) для вставки AgeI, и 5’-GTTTCTGGCGTCGACTCTTTCGGCACGCTGAACTTCACCTCTGACTTCCAGGAAC-3’ (SEQ ID NO:110) и 5’-CGAAAGAGTCGACGCCAGAAACGCCCGTGTAGTAGTTA CGACGTCCCGCGCG-3’ (SEQ ID NO:111) для вставки SalI в ген NifD из конструкции pTopoH-J (Пример 4). Этот фрагмент от AgeI до SalI был кодон-оптимизирован для экспрессии N. benthamiana. Полученный вектор расщепляли SbfI и лигировали с B-ori, расщепленным SbfI, создавая вектор положительного контроля, обозначенный pSO043, кодирующий NifD дикого типа, а также другие полипептиды Nif.
Область AgeI-SalI NifD, содержащую каждую из аминокислотных замен, амплифицировали с помощью PCR, чтобы добавить сайты рестрикции AgeI и SalI в те же позиции, что и в pSO043. Для этого использовали праймеры 5’-GACCAATGCTACCGGTGAGAGGAACC-3’ (SEQ ID NO:112) и 5’-GTTAAGAGTCCCGAAAGAGTCGACACCAG-3’ (SEQ ID NO:113), и DNA из конструкций SN114, SN118, SN119, SN120, SN123, SN124 и SN125 в качестве матрицы, каждая из которых кодирует свой вариант последовательности NifD. Затем амплифицированные фрагменты варианта AgeI-SalI NifD лигировали в pSO043, расщепленную AgeI и SalI, в результате чего получали ряд конструкций, обозначенных pSO044-050. Таким образом, эти конструкции содержали область AgeI-SalI, которая была кодон-оптимизирована для экспрессии в растительных клетках, тогда как остальная часть гена NifD была кодон-оптимизирована для экспрессии в E. coli. Два других вектора NifD также были сконструированы аналогичным образом с использованием SN100 (NifD-Var13, имеющей аминокислотные остатки 99 и 103, замещенные пятью остатками аланина) и EC38014, имеющей ген NifD с кодон-оптимизированной N. benthamiana, в качестве DNA-матриц, что привело к образованию pSO052 и pSO053, соответственно.
Конструкции бактериальной экспрессии, имеющие варианты генов NifD, вводили в штамм JM109 E. coli вдоль вектора индукции экспрессии pN249 и исследовали на функцию нитрогеназы с использованием анализа восстановления ацетилена (ARA). Бактерии, трансформированные совместно с pSO053 (позитивная регуляцияи, NifD дикого типа) и вариантами NifD, кодированными pSO052 (замещения аланином остатков от 99 до 103), pSO044 (Y100Q), pSO045 (NYY99-101HKG), pSO046 (Y100K) и pSO047 (YY100-101KA), причем каждый в некоторой степени продуцировал этилен. Количество этилена, продуцированного pSO044, pSO045, pSO046 и pSO047, составило 147%, 33%, 94% и 67%, соответственно, по сравнению с позитивной регуляцией, а pSO052 также продуцировал этилен в количестве 14% по сравнению с позитивной регуляцией. Однако клетки E. coli, содержащие pSO048, pSO049 и pSO050 и все с заменой R98K, продуцировали только следовые количества этилена со скоростью, большей, по сравнению с негативной регуляцией, что указывает на то, что эти мутанты NifD были почти неактивны для нитрогеназы. Аналогичным образом конструкция, имеющая двойную замену YY100-101QT, давала 107% активность ARA по сравнению с контрольной NifD дикого типа (Таблица 10). Следовательно, замены Y100Q и YY100-101QT давали повышенную активность NifD по сравнению с последовательностью NifD дикого типа. Был сделан вывод, что аргинин в позиции 98 необходим для функции NifD, что согласуется с его полной консервацией в природных последовательностях NifD, в которых можно предположить активность.
В более общем плане был сделан вывод, что были идентифицированы варианты NifD, которые сохраняли существенную функцию NifD, действительно полную или даже повышенную функцию NifD для некоторых вариантов, которые не подвергались вторичному расщеплению, наблюдаемому в последовательности NifD K. oxytoca дикого типа. Также был сделан вывод о том, что устойчивость полипептидов NifD к вторичному расщеплению в митохондриях растения сочетается с повышенной активностью нитрогеназы, что последняя продемонстрирована для модифицированной последовательности NifD в бактериальной системе. Также был сделан вывод, что можно идентифицировать другие варианты NifD, которые не подвергались вторичному расщеплению, но которые утратили функцию NifD.
Пример 10. Прочие полипептиды NifD
Моделирование структуры NifD-NifK вокруг сайта вторичного расщепления в NifD
Белковая структура полипептида NifD из K. oxytoca, представленная в PDB: 1QGU визуализировали с помощью программного обеспечения PyMOL, в частности обращая особое внимание на структуре вокруг сайта вторичного расщепления, когда NifD связывался с полипептидом NifK из K. oxytoca. Было обнаружено, что сайт вторичного расщепления локализуется на границе раздела полипептидов NifD и NifK, внутри комплекса, и в непосредственной близости от основного FeMo-кофактора (Рисунок 11). В состоянии покоя остаток Arg97 NifD координировался с мостиковым сульфидным лигандом (S5), локализованным между Fe3 и Fe7 FeMo-ко, что объясняет, почему Arg97 был полностью консервативен в функциональных полипептидах NifD. Считалось, что он играет важную роль в стабилизации отрицательных зарядов более укороченного края кластера (Fe1-Fe3-Fe7) (Spatzal et al., 2016). Гидроксильная группа Tyr100 в NifD образовывала водородные связи с аминогруппой в Arg98 в NifD, гидроксильной группой в Ser515 в NifK и карбоксильной группой Asp517 в NifK из K. oxytoca, что также показывает ее важность для функции NifD.
Гомологические модели для вариантов NifD, содержащих замены Y100Q и Y100K, были подготовлены с использованием сервера SWISS-MODEL (Waterhouse et al., 2018). Последовательность NifD Y100Q или NifD Y100K использовали в качестве последовательности-мишени. Последовательность NifK из K. oxytoca снова была добавлена к модели в качестве гетеромишени. Для варианта Y100Q модель предсказала, что аминогруппа Gln100 полипептида NifD образует водородную связь с карбоксильной группой Asp517 в NifK и остовом атома карбонильного кислорода Tyr514 в NifK. Для варианта Y100K модель предсказала, что аминогруппа Lys100 также образует водородные связи с Asp517 и Tyr514 в NifK. Взаимодействие Tyr100 с Ser515 в NifK было заменено взаимодействием Gln100 или Lys100 с остовом атома кислорода Tyr514 в NifK. Эти наблюдения согласуются с сохранением активности NifD для замен Y100Q и Y100K.
Наблюдения, что (i) последовательность вокруг сайта вторичного расщепления была внутренней в полипептиде NifD, когда он уложен в его активной конформации, и (ii) что полипептид NifD-линкер-NifK, включающий последовательность NifD K. oxytoca дикого типа, был расщеплен, позволяли предположить что вторичное расщепление происходило, когда полипептид был разложен или раскладывался в митохондриях.
Филогенетический анализ, описанный выше (Пример 8), показывает, что аминокислотные остатки лейцина, фенилаланина, метионина и глутаминовой кислоты также представлены в природных полипептидах NifD в позиции, соответствующей Y100 в NifD K. oxytoca, то есть в позиции +1 относительно вторичного сайта расщепления и эти полипептиды NifD считались функциональными. В этих полипептидах за аминокислотой Leu в позиции, соответствующей аминокислоте 100, в позиции 101 следовали аланин (53 последовательности), метионин (41 последовательность), валин (10 последовательностей), треонин (4 последовательности), фенилаланин (4 последовательности) или тирозин (2 последовательности). Когда аминокислота, соответствующая позиции 100, представляла собой Phe, то следующей аминокислотой обычно был аланин (23 последовательности), а в некоторых случаях был серин (2 последовательности) или тирозин (2 последовательности). За Met в позиции 100 следовали аланин (3 последовательности), метионин (3 последовательности), глицин (2 последовательности), валин (1 последовательность) или треонин. За Glu в позиции 100 следовал треонин (1 последовательность). Однако наличие Phe, Leu или Met в позиции 100 не создавало водородные связи, которые Y100 имел с аминокислотами Ser515 и Asp517 в NifK из K. oxytoca.
Для исследования их функции, генетические конструкции, содержащие замены Y100L, Y100F и Y100M, были введены в последовательность NifD K. oxytoca аналогично заменам, описанным выше. Эти конструкции, кодирующие варианты NifD, исследовали на фенотип вторичного расщепления после введения в клетки листьев N. benthamiana и на функцию NifD в E. coli с использованием системы pMIT2.1, аналогично тому, как описано в Примере 8. Все три из этих полипептидов NifD, имеющих замены Y100L, Y100F и Y100M, также подвергались вторичному расщеплению, показывая, что аминокислотные последовательности в этом сайте все еще распознаются MPP в митохондриях растения. Остальные 14 возможных замен в позиции 100 легко исследуются аналогичным образом.
Прогнозирование и исследование на расщепление природных последовательностей NifD
На основе мутационного и филогенетического анализов были сделаны прогнозы относительно различных природных последовательностей NifD, а именно относительно их расщепления или отсутствия расщепления, или меньшего расщепления в области, соответствующей аминокислотам 97-102 из NifD (SEQ ID NO:18) K. oxytoca. Чтобы исследовать эти прогнозы, была выбрана одна последовательность из каждого из трех кластеров последовательностей NifD, которые содержали наибольшее количество членов, имеющих последовательность RRNYY (SEQ ID NO:102), которые, по прогнозам, должны расщепляться. Эти выбранные последовательности NifD были из Azotobacter vinelandii, Azospirillum brasilense и Sinorhizobium fredii. Эти последовательности обозначены как SEQ ID NO:148-150. Были идентифицированы три другие аминокислотные последовательности NifD, которые не имели последовательности RRNY (SEQ ID NO:101), а вместо этого имели RRNQ (SEQ ID NO:103), RRNK (SEQ ID NO:104) или RRFK (SEQ ID NO:106) в соответствующей последовательности (Таблица 13). Эти выбранные последовательности NifD были из Clorobium tepidum, Desulfotomaculum ferrireducens и Desulfovibrio vulgaris и содержали остаток глутамина или лизина, а не тирозин в эквиваленте позиции 100. Эти последовательности обозначены как SEQ ID NO:151-153. Было предсказано, что эти три полипептида будут менее подвержены расщеплению внутри этих последовательностей.
Эти выбранные последовательности были выровнены с последовательностью K. oxytoca с использованием инструмента Emboss Needle Pairwise Alignment Tool для определения степени идентичности, что также показано в Таблице 13. Было отмечено, что SEQ ID NO:151-153, не содержащие последовательности RRNY (SEQ ID NO:101), менее чем на 40% идентичны SEQ ID NO:18.
Для исследования прогноза по каждому случаю вторичного расщепления, были созданы генетические конструкции (SN221-226), кодирующие слитые полипептиды MTP-FAγ51::NifD::HA, где последовательность NifD была такой же, как природная последовательность, для введения в листья N. benthamiana. Белковые экстракты получали через 5 дней после инфильтрирования и подвергали электрофорезу SDS-PAGE и анализу вестерн-блот. Было обнаружено, что слитые полипептиды, содержащие последовательность RRNYY (SEQ ID NO:102) из NifD A. vinelandii и S. fredii, показывали интенсивные полосы вторичного расщепления в ходе анализа вестерн-блот, причем более 50% полипептидов расщеплялись на вторичном сайте, тогда как полипептид NifD из D. vulgaris, не имеющий RRNYY (SEQ ID NO:102), показал небольшое вторичное расщепление. Полипептиды C. tepidum и D. ferrireducens показали некоторое вторичное расщепление, но меньшее, чем у NifD из A. vinelandii и S. fredii.
Эксперимент повторили, включая генетические конструкции в качестве контроля, которые кодировали слитые полипептиды, и которые не могли быть расщеплены с помощью MPP из-за замен аланина в последовательности MTP-FAγ51 (дорожки, отмеченные A на Рисунке 12), или которые не имеют последовательности MTP, но имеют мотив 6xHis вместо этого (дорожки, отмеченные буквой C), равные по размеру формам, процессированным MPP. Анализ вестерн-блот (Рисунок 12) снова показал, что соотношение полноразмерного NifD и продукта вторичного расщепления менялось для всех шести слитых полипептидов NifD. В работе Burén et al. (2017B) приводили данные о наличии в митохондриях дрожжей продукта деградации NifD из A. vinelandii массой прим. 48 кДа. Размер этого полипептида соответствовал предсказанному вторичному расщеплению MPP в сайте RRNYY (SEQ ID NO:102). В повторном эксперименте продукт деградации такого размера наблюдали для полипептида NifD A. vinelandii. Также наблюдался второй продукт деградации с молекулярной массой приблизительно равной 40 кДа, что позволяет предположить наличие скрытого сайта вторичного расщепления. Для генетической конструкции, кодирующей локализованный в цитоплазме слитой полипептид 6xHis::NifD::HA не наблюдались два продукта деградации, что указывает на то, что второй продукт деградации также возник в результате активности митохондриальной протеазы. Также было отмечено различие между процессингом в митохондриях растения по сравнению с митохондриями дрожжей. Необходимы дальнейшие исследования для определения происхождения второго продукта деградации слитого полипептида AvNifD A. vinelandii в растительных клетках.
Слитой полипептид, включающий последовательность NifD из A. brasilense, в основном присутствовал в непроцессированной форме, что указывает на низкую эффективность импорта слитого полипептида AbNifD в митохондрии. По этой причине для этого было трудно оценить степень расщепления MPP. Важно отметить, что на этот раз у слитого полипептида, включающего последовательность SfNifD из S. fredii, наблюдался только слабый продукт деградации.
Относительное содержание полипептидов MTP51::NifD::HA сильно менялось, хотя во всех генетических конструкциях использовался один и тот же промотор. Содержание слитых полипептидов NifD из A. brasilense, A. vinelandii и D. vulgaris было низким по сравнению с слитыми полипептидами NifD из C. tepidum и S. fredii.
Эксперимент расширили путем фракционирования белковых экстрактов на растворимые и нерастворимые фракции, как описано в Примере 1. Было обнаружено, что слитые полипептиды NifD из C. tepidum, D. ferrireducens и S. fredii были в некоторой степени растворимы, примерно до 50% для NifD из D. ferrireducens.
Был сделан вывод, что природные последовательности NifD, имеющие последовательность RRNYY (SEQ ID NO:102), менее предпочтительны из-за их тенденции к вторичному расщеплению внутри этой последовательности в митохондриях растения, но есть и исключения, например, NifD из S. fredii.
Таблица 13. Последовательности полипептида NifD дикого типа в последовательности, соответствующей аминокислотам 97-102 K. oxytoca. Показан % идентичности аминокислотной последовательности SEQ ID NO:18. Было предсказано, что первые три последовательности, содержащие RRNYY (SEQ ID NO:102), будут расщепляться в этой области, тогда как последние три последовательности не будут в ней расщепляться.
(SEQ ID NO:102)
(SEQ ID NO:102)
(SEQ ID NO:102)
(SEQ ID NO:105)
(SEQ ID NO:105)
(SEQ ID NO:107)
Пример 11. Варианты NifD в окружении слитых полипептидов NifD-NifK
Также исследовали влияние замены Y100Q на процессинг и функцию NifD в окружении слитого полипептида NifD-линкер-NifK. Для этого сначала модифицировали вектор pMIT2.1 для трансляционного слияния других кодирующих областей NifD и NifK дикого типа, как показано ниже. Структура оперона между генами NifD и NifK в pMIT2.1, кодирующими отдельные полипептиды NifD и NifK, была заменена нуклеотидной последовательностью для обеспечения трансляционного слияния полипептидов NifD и NifK, соединенных линкером из 30 аминокислот (ATPPPGSTTTAYPYDVPDYATPPPGSTTTA, SEQ ID NO:116), который включал метку эпитопа НА (YPYDVPDYA, SEQ ID NO:115). Фрагмент DNA, кодирующий этот полипептид NifD::linker(HA)::NifK, происходил из гена NifD::FLAGlinker::NifKs (Allen et al., 2017), за исключением того, что нуклеотидная последовательность, кодирующая аминокислоты эпитопа FLAG, была заменена последовательностью, кодирующей эпитоп НА, с образованием вектора, обозначенного здесь как pTopoH-J-DHAK. Затем вторую немодифицированную половину pMIT2.1 (NifB-ori), расщепленную SbfI, лигировали с pTopoH-J-DHAK после расщепления SbfI, и в результате получали pSO018. Таким образом, эта конструкция кодировала трансляционное слияние NifD::linker(HA)::NifK со всеми другими генами Nif, как в pMIT2.1, а аминокислотная последовательность NifD оставалась немодифицированой по сравнению с последовательностью K. oxytoca дикого типа.
Введение мутации Y100Q в кодирующую область NifD в pTopoH-J и pTopoH-J-DHAK достигалось с помощью мутагенеза с использованием праймеров 5’-GTCGTAACCAATACACGGGCGTTTCTGGCGTCGACTCTTTCGGCACG-3’ (SEQ ID NO:117) и 5’-GCCCGTGTATTGGTTACGACGTCCCGCGCGAGAG TACTGGC-3’ (SEQ ID NO:118) для осуществления нуклеотидных замен T298C и C300A, и заменой кодона ТАС тирозина (Y) на кодон САА глутамина (Q). Полученные векторы pTopoH-J, кодирующие неслитой или слитой NifD (Y100Q), были расщеплены SbfI и лигированы NifB-ori, также расщепленным SbfI, с образованием pSO054, который был преобразованным pMIT2.1, кодирующим NifD (Y100Q), и образованием pSO055, который был преобразованным pMIT2.1, кодирующим полипептид трансляционного слияния NifD(Y100Q)::linker(HA)::NifK.
Эти генетические конструкции исследовали с использованием анализа восстановления ацетилена в E. coli. Конструкция pSO054 (кодирующая неслитой NifD (Y100Q)) и pSO055 (кодирующая слитой NifD(Y100Q)::linker(HA)::NifK) продуцировали этилен в количестве от 80% до 90% по сравнению с их соответствующими конструкциями pSO005 и pSO018 позитивной регуляции. Это показало, что мутация Y100Q не понижала активность NifD в окружении слитого полипептида NifD::linker::NifK, при этом активность снижалась лишь незначительно.
Пример 12. Растворимость NifD дикого типа и варианты последовательностей в митохондриях растения
В Примере 3 описываются эксперименты, показывающие, что многие полипептиды Nif, экспрессированные в форме слитого полипептида MTP::Nif для локализации в митохондриях, были по существу нерастворимы или только умеренно растворимы при экспрессии в виде одного полипептида. Данные также продемонстрировали, что процесс нацеливания слитых полипептидов Nif на митохондрии или саму митохондриальную среду, или на то и другое вместе, отрицательно влияет на растворимость полипептида Nif, по меньшей мере, для NifD, NifH и NifK по сравнению с локализацией в цитоплазме. Поскольку растворимость белковых компонентов нитрогеназы в митохондриальном матриксе считается предпосылкой для функционального восстановления нитрогеназы в митохондриях эукариотических клеток, изобретатели стремились определить причины растворимости полипептида Nif. В частности, учитывая важность NifD, NifK и NifH, были исследованы несколько концепций для увеличения растворимости этих важнейших полипептидов, как описано ниже. Нерастворимость в митохондриях могла быть следствием неправильной укладки белка, неправильного гликозилирования или другой посттрансляционной модификации, образования агрегатов или ассоциации с клеточными мембранами или сочетания этих или других причин.
Растворимость слитых полипептидов NifD - влияние промотора, MTP и последовательностей NifD
Первоначально набор экспрессируемых в растении полипептидов MTP::NifD исследовали на растворимость, включая способность N- и C-концевых модификаций влиять на растворимость форм процессированных и непроцессированных MPP. Для этой цели ряд конструкций MTP::NifD, включая некоторые из описанных выше, были инфильтрированы в листья N. benthamiana с помощью A. tumefaciens (Таблица 14). Эти конструкции различались по промотору для экспрессии (промоторы e35S или SCSV S4) или по кодируемым полипептидным последовательностям MTP или NifD (расщепленным или не расщепленным на вторичном сайте). Все они содержали метку эпитопа HA, слитую с C-концом полипептида NifD, за исключением полипептида, кодируемого SN75, который имеет последовательность эпитопа HA, слитую с каждым N-концом и C-концом NifD, таким образом фланкируя полипептид NifD. В качестве позитивной регуляции для растворимого полипептида NifD также инфильтрировали генетическую конструкцию SN33 (Пример 3), кодирующую немитохондриально нацеленную версию NifD. В каждом случае, конструкция SN46, кодирующая MTP-Su9::NifK, подвергалась совместному инфильтрированию с конструкцией NifD для увеличения накопления NifD. Для каждого инфильтрирования белки экстрагировали из каждой пятнистости листьев и фракционировали на растворимые и нерастворимые фракции, как описано в Примере 1, а также оставляли некоторые нефракционированные образцы («общий белок»). Образцы анализировали путем загрузки их в соседние дорожки для электрофореза SDS-PAGE и анализа вестерн-блот с использованием анти-HA для обнаружения MPP-процессированных и непроцессированных полипептидов MTP::NifD::HA и MTP::HA::NifD::HA.
По данным анализа вестерн-блот, продуцированный из SN33 и митохондриально не нацеленный полипептид HA::NifD был почти полностью растворим (оценка растворимости 4, Таблица 14). Напротив, полипептид MTP-FAγ::NifD::HA, кодированный SN10, и его MPP-процессированное производное, либо не обнаруживались, либо едва обнаруживались в растворимой фракции, поэтому были практически нерастворимыми. Модификация промотора путем замены промотора SCSV S4 (SN06) на промотор e35S, по-видимому, несколько увеличивает содержание растворимого полипептида NifD::HA. Изменение MTP путем замены последовательности FAγ51 последовательностями CPN60 или Su9 MTP (SN04, SN14) не привело к заметному увеличению растворимости NifD. Небольшое увеличение растворимости наблюдалось, когда аминокислотная замена Y100Q была включена в аминокислотную последовательность NifD (SN114). Ни одна из этих модификаций не дала большого эффекта. Однако резкое изменение как уровня экспрессии NifD, так и растворимости произошло с SN75. По меньшей мере, 50% слитого полипептида, кодированного SN75, содержащего метку эпитопа НА между MTP-FAγ51 и последовательностью NifD, а также вторую, С-концевую метку эпитопа НА, приходились на растворимую фракцию. Любопытно, что другой N-концевой эпитоп, локализованный между MTP и NifD, дал другой результат, а именно конструкция (SN19), кодирующая полипептид MTP-CoxIV::TwinStrep::NifD:HA, давала в основном нерастворимый полипептид NifD.
Принимая во внимание результат с SN75, была создана аналогичная конструкция (SN140) для экспрессии NifK, имеющая метку эпитопа HA, связанную с Gly-Gly, помещенную между последовательностями MTP-FAγ51 (SEQ ID NO:36) и NifK. После инфильтрирования SN140 в клетки листьев N. benthamiana получали фракции растворимого, нерастворимого и общего белка. Однако, в отличие от SN75, электрофорез SDS-PAGE и анализ вестерн-блот показали, что слитой полипептид NifK оставался нерастворимым. Этот результат продемонстрировал, что встраивание HA линкер-GG в слитой полипептид оказывает различное влияние на растворимость белка в зависимости от его полипептида Nif, в данном случае NifD относительно NifK.
В целом, эти результаты подтвердили, что процесс нацеливания полипептидов NifD на митохондрии или саму митохондриальную среду отрицательно влияет на растворимость NifD. Они также показали, что модификация N-конца может решить эту проблему, по меньшей мере частично.
Таблица 14. Растворимость слитых полипептидов MTP::NifD после экспрессии в растительных клетках и процессинга MTP с помощью MPP, определенная с помощью электрофореза SDS-PAGE и анализа вестерн-блот растворимых и нерастворимых фракций. Показатели растворимости приведены для каждого слитого полипептида: 0, растворимый полноразмерный полипептид NifD не обнаружен; 1, только что обнаруженный растворимый полипептид; 2, растворимый полипептид обнаружен, но <50% от общего полипептида; 3, растворимый полипептид, обнаружен, но > 50% от общего полипептида; 4, по меньшей мере, 90% от всего полипептида представляет собой растворимый полипептид.
3’HA
Растворимость слитых полипептидов NifD::линкер::NifK
Учитывая эти результаты, было исследовано влияние на растворимость NifD другого С-концевого продолжения, а именно добавления последовательности NifK для обеспечения трансляционного слияния MTP::NifD::линкер(HA)::NifK (Allen et al., 2017). Для этого создали генетическую конструкцию SN68, которая, как и SN10, содержала: сильный промотор e35S для экспрессии, и область TMV-омега 5’-UTR для эффективной трансляции (Gallie et al., 1987). SN68 кодировала слитой полипептид, который содержал MTP-FAγ51 с линкером Gly-Gly, слитым с N-концом NifD, затем линкер из 30 аминокислот, содержащий последовательность метки эпитопа НА, которая ранее использовалась в pRA20, за которой следовала последовательность NifK. Это показано схематически на Рисунке 13. Аминокислотная последовательность NifD соответствовала SEQ ID NO:18. Для экспрессии в N. Benthamiana кодирующая белок область была кодон-оптимизирована.
Этот полипептид исследовали на растворимость путем инфильтрирования SN68 в N. benthamiana и выделения фракций растворимого, нерастворимого и общего белка. Белковые фракции подвергали электрофорезу SDS-PAGE и анализу вестерн-блот. На мембранах для блоттинга появились две полосы (Рисунок 14), которые были немного меньше, чем ожидалось, и, вероятно, представляли собой расщепление на сайте вторичного расщепления в последовательности NifD. Однако, несмотря на это, было отмечено, что большинство слитых полипептидов, содержащих эпитоп НА и последовательность NifK, находилось в растворимой фракции и лишь небольшое количество находилось в нерастворимой фракции (Рисунок 14). Это был первый случай, когда изобретатели наблюдали в основном растворимый полипептид NifK.
Поскольку полипептид, кодированный SN68, включал аминокислотную последовательность, которая была восприимчива к вторичному расщеплению между аминокислотами 97-102 NifD, то была создана вторая соответствующая конструкция, содержащая аминокислотную замену Y100Q, которая показала защиту NifD от вторичного расщепления в митохондриях (Пример 6). Генетическую конструкцию обозначили SN159. Чтобы различить процессированные и непроцессированные слитые полипептиды при электрофорезе SDS-PAGE и тем самым установить, расщеплялся ли слитой полипептид, кодированный SN159, с помощью MPP в последовательности MTP, была создана третья конструкция, идентичная SN159, за исключением того, что последовательность MTP-FAγ51 была модифицирована заменами аланина, которые сделали бы ее устойчивой к процессингу митохондрий с помощью MPP. Были сделаны те же замены аланина в MTP, что и в полипептиде конструкции SN66. Таким образом, третья конструкция SN160 была сконструирована для получения слитого полипептида mMTP::NifD::линкер(HA)::NifK, который не будет процессироваться MPP и, следовательно, давать продукт большего размера, чем процессированный продукт из SN159. Кроме того, была изготовлена четвертая, контрольная конструкция, обозначенная SN176, кодирующая слитой полипептид, в котором отсутствует последовательность MTP и, следовательно, не нацелен на митохондрии, а скорее будет локализован в цитоплазме. Для этой конструкции последовательность MTP-FAγ51 из SN159 была заменена меткой 6xHis, связанной двумя глицинами с инициирующим кодоном NifD. Последовательность 6xHis+Gly-Gly была очень похожа по размеру на последовательность scar, которая, как предполагалось, будет продуцироваться из MTP-FAγ51 после MPP-процессинга. Было предсказано, что если SN159 была процессирована правильно, то белковые продукты из SN176 и SN159 будут иметь практически одинаковую длину (1040 остатков/116 251 Da для SN176 и 1042 остатка/116 317 Da для процессированной SN159).
Эти конструкции SN68, SN159, SN160 и SN176 инфильтрировали отдельно в листья N. benthamiana, и через 5 дней получали три белковые фракции из каждой инфильтрованной области листа: общий белок, растворимый белок и нерастворимый белок, как описано в Примерах 1 и 3. Фракции анализировали электрофорезом SDS-PAGE и анализом вестерн-блот с антителом HA. Электрофорез SDS-PAGE проводили дольше, чем обычно, ввиду больших размеров полипептидов, чтобы обеспечить лучшее разрешение.
Конструкции SN159 и SN160 давали отличающуюся полосу полипептида, имеющую молекулярную массу приблизительно 120 кДа, причем основной полипептид из SN160 был заметно больше, чем у SN159. Полипептид из SN159 оказался того же размера, что и полипептид из SN176, в котором отсутствует последовательность MTP. Из этого был сделан вывод, что полипептид, продуцированный из SN159, был эффективно процессирован MPP. Напротив, полипептиды, продуцированные из SN68, были меньше по размеру и поэтому предполагалось, что они включают продукт вторичного расщепления в последовательности NifD. Было предсказано, что полипептид, полученный из SN68 без замены Y100Q, будет подвергаться вторичному расщеплению и, следовательно, продуцировать продукт из 933 остатков/104 403 Da. Наблюдалась полипептидная полоса такого размера.
Наиболее приятным и удивительным для изобретателей стал результат анализа растворимости полипептидов, продуцированных из этих конструкций. В растворимой фракции наблюдалось больше полипептида, продуцированного из SN159, чем в нерастворимой фракции. Это был первый случай, когда изобретатели увидели это для митохондриально нацеленного полипептида NifD. Более того, было показано, что процессированный полипептид MTP::NifD::линкер(HA)::NifK действует в отношении NifD и NifK в системе бактериального анализа (Пример 11). Таким образом, авторы изобретения пришли к выводу, что они успешно модифицировали полипептиды Nif для продуцирования растворимого функционального полипептида, имеющего функции как NifD, так и NifK, который был устойчивым к вторичному расщеплению последовательности NifD в последовательности RRNYY (SEQ ID NO:102).
Помимо растворимости, имелись некоторые важные различия в процессинге между отдельно экспрессированными полипептидами NifD и NifD::линкер::NifK. Во-первых, в отличие от полипептида NifD из SN10 и его вариантов замещения (Пример 6), полипептид MTP::NifD::линкер::NifK, содержащий последовательность NifD дикого типа (SN68), полностью процессировался с помощью MPP. Даже несмотря на то, что продукт вторичного расщепления массой ~ 48 кДа преобладал из конструкции SN10 и некоторых других конструкций вариантов NifD, всегда обнаруживали полноразмерный полипептид NifD, который не был расщеплен на сайте RRNYY (SEQ ID NO:102). Во-вторых, несмотря на использование одного и того же MTP-FAγ51 для SN159, SN10 и других вариантов NifD, процессинг с помощью MPP оказался полным для SN159, тогда как процессированный так и непроцессированный MTP-FAγ51::NifD наблюдался всегда, когда NifD экспрессировался в виде отдельного полипептида. Следовательно, случайно, слитой полипептид из SN159 не только обладал устойчивостью к вторичному расщеплению в митохондриях и был преимущественно растворим, но также оказался полностью процессированным в каноническом сайте в последовательности MTP.
Выделение слитых полипептидов NifD::линкер::NifK
Слитой полипептид NifD::линкер(HA)::NifK, кодированный SN159, был выделен из образцов листьев N. benthamiana следующим иммуно-селективным способом. Двенадцать кусочков листьев, каждый размером примерно 2 см2 и которые были инфильтрированы SN159, измельчали в 10 мл буфера для определения растворимости. Буфер для определения растворимости содержал: 100 ммоль Трис pH 8,0, 150 ммоль NaCl, 0,25 ммоль/л маннита, 5% (об./ об.) глицерина, 1% (мас./об.) PVP40, 0,1% (об./об.) Tween 20, 2 ммоль TCEP, 0,2 ммоль PMSF и 10 мкмоль/л леупептина. Ожидалось, что использование низкого уровня детергента (0,1% Tween20) приведет к экстракции только растворимых белков. Измельченную смесь центрифугировали при 5500 x g в течение 15 минут при 4 °C, а надосадочную жидкость переносили в чистую пробирку. Магнитные микроносители агарозы (Sigma) анти-НА один раз промывали буфером, содержащим 50 ммоль Трис pH 8,0 и 75 ммоль NaCl (буфер TN), а затем магнитные микроносители добавляли к надосадочной жидкости для образования иммунопреципитата полипептидов, имеющих эпитоп НА. Смесь инкубировали в течение 1 ч при 4°C при медленном вращении и давали осесть магнитным микроносителям. Образец надосадочной жидкости сохраняли как «несвязанные белки». Магнитные микроносители промывали 5 раз в 1 мл буфера TN каждый раз, центрифугируя каждый раз для осаждения магнитных микроносителей при 1000 x g в течение 2 минут и при комнатной температуре. Наконец, к магнитным микроносителям добавляли 60 мкл буфера Лэммли и смесь нагревали при 95°C в течение 5 минут, чтобы высвободить связанные белки и денатурировать их. Дубликаты образцов загружали для электрофореза SDS-PAGE.
Один из гелей наносили на мембрану и обрабатывали как вестерн-блот. Наблюдалась интенсивная полипептидная полоса с размером, ожидаемым для MPP-процессированного полипептида NifD::линкер(HA)::NifK, а также две менее интенсивные полосы для полипептидов меньших размеров, которые считались продуктами деградации, вероятно, вызванные расщеплением протеазой на скрытых сайтах в NifD. Поскольку последовательность NifD в полипептиде имела аминокислотную замену Y100Q, то маловероятно, что дальнейшее расщепление/деградация протеазой происходила в этом сайте в NifD, а скорее в одном или нескольких новых сайтах. Анализ вестерн-блот также показал две интенсивные полосы, которые, как предполагалось, представляют собой полипептиды мышиного Ig массой 50 кДа и 25 кДа, присутствующие на магнитных микроносителях агарозы анти-НА, которые использовались для образования иммунопреципитата.
Второй гель для электрофореза SDS-PAGE окрашивали красителем Кумасси и использовали для изготовления срезов геля в областях, соответствующих полосе NifD::линкер(HA)::NifK (Образец 1) и более мелким продуктам деградации (Образец 2). Белки в этих гелевых срезах расщепляли трипсином и анализировали способом LC-MS, как описано в Примере 1. Экстрагированные триптические пептиды сушили и повторно суспендировали в 30 мкл 1% муравьиной кислоты. Первоначально 5 мкл триптических пептидов из каждого из гидролизатов вводили в систему 6600 Triple TOF MS с использованием микро-HPLC Eksigent. (55 мин). Остаточные триптические пептиды хранили при -20 °C.
Данные обрабатывались в ПО ProteinPilot на основе видоспецифичных баз данных UniProt Knowledgebase (UniProtKB), к которым добавлялись пользовательские базы данных и дополнительные базы данных: Uniprot-Nbenth+Пользовательская база данных Nif+Общий репозиторий дополнительных белков. Несколько специфичных пептидов из целевого полипептида были положительно идентифицированы в Образце 1, причем 2 пептида из последовательности NifD идентифицированы с уровнем достоверности > 95%, а еще один пептид из NifD и два других пептида в последовательности NifK были идентифицированы с уровнем достоверности, составившим 94,9, 93,3 и 55,3%. Два пептида scar, полученные в результате расщепления MPP в последовательности MTP, ISTQVVR (SEQ ID NO:119) и SISTQVVR (SEQ ID NO:120), не были обнаружены в базе данных, но были обнаружены с помощью более чувствительного целевого спектрометра MRM 6500 Q-trap с 6 транзициями ион/пептид и со временем удержания 2,83 мин и 3,15 мин, соответственно. Оценка доминирующих транзиций ионов (+ 2y6) показала, что пептид SISTQVVR (SEQ ID NO:120) был немного более распространенным, чем ISTQVVR (SEQ ID NO:119) в Образце 1.
Был сделан вывод, что Образец 1 действительно содержал MPP-процессированный полипептид NifD::линкер(HA)::NifK и что полипептид был экстрагирован из клеток N. benthamiana в растворимой форме.
Образец 2 имел более низкое содержание белка, поэтому анализ был затруднен. Тем не менее, один триптический пептид был идентифицирован внутри последовательности NifD и свидетельствовал о наличии второго пептида внутри NifD. Два пептида scar ISTQVVR (SEQ ID NO:119) и SISTQVVR (SEQ ID NO:120) не были обнаружены в базе данных и более чувствительным спектрометром MRM 650 Q-trap. Эти данные согласуются с полипептидами из Образца 2, возникшими в результате дополнительного расщепления в последовательности NifD.
Усиление растворимости полипептида NifK
Авторы изобретения исследовали, способна ли совместная экспрессия полипептидов NifD и NifK из отдельных конструкций усиливать растворимость полипептида NifK по сравнению с экспрессией NifK без NifD. Описанные выше эксперименты с SN140 (MTP-HA::NifK) показали, что полипептид при отдельной экспрессии был практически нерастворим. Листья N. benthamiana были инфильтрированы смесью штаммов A. tumefaciens, отдельно трансформированных с SN140 и либо SN10, SN114, либо SN117. SN10 кодировала последовательность NifD дикого типа, тогда как SN114 и SN117 содержали аминокислотные замены в NifD для уменьшения вторичного расщепления. Белковые экстракты, содержащие растворимые и нерастворимые фракции, а также нефракционированные белки, подвергали электрофорезу SDS-PAGE и анализу вестерн-блот, как и ранее.
Мембраны для блоттинга показали, что имело место значительное увеличение растворимости слитого полипептида NifK, экспрессированного из SN140, при совместном введении с любой из конструкций NifD. В отсутствие NifD, NifK практически не обнаруживался в растворимой фракции, тогда как в присутствии NifD примерно равные количества NifK были обнаружены в растворимой и нерастворимой фракциях. Был сделан вывод, что растворимость полипептида MTP::NifK увеличивалась за счет совместной экспрессии NifD вместе в одной и той же растительной клетке, даже при экспрессии в виде отдельных полипептидов. Это добавило к описанному выше наблюдению, что слитой полипептид MTP-NifD-линкер-NifK дает более растворимую форму полипептида NifK. Был также сделан вывод о том, что оба наблюдения указывают на ассоциацию полипептидов NifD и NifK в митохондриальном матриксе, обязательно для слитого полипептида NifD-линкер-NifK, но также при экспрессии в виде отдельных полипептидов.
Пример 13. Очистка митохондрий растения с использованием слитого белка и магнитных микроносителей
Авторы изобретения разработали способ быстрой очистки митохондрий растения, чтобы лучше исследовать локализацию и функцию экзогенных полипептидов в митохондриях, например, полипептидов Nif, которые они хотели ввести в эту субклеточную органеллу. Традиционные способы выделения высокообогащенных митохондрий растения обычно требуют процессинга свежесобранного листового материала различными буферами с дальнейшей последовательностью стадий центрифугирования для удаления немитохондриальных компонентов (Millar et al., 2007). Эти способы требовали значительных количеств исходного материала (напр., от 20 до 40 граммов растительного материала), а весь процесс занимал много часов, прежде чем очищенные митохондрии были готовы к использованию или анализу. Были разработаны более быстрые способы выделения с меньшим количеством растительного материала (Millar et al., 2007). Однако эти способы лучше всего рассматривать как обогащение митохондрий, поскольку продукты обычно все еще содержат другие клеточные компоненты (Carrari et al., 2003).
В описанных здесь анализах листьев N. benthamiana могут быть применены к одному листу от 8 до 10 или более «зон инфильтрирования», при этом каждая зона способна экспрессировать один или несколько (до примерно 8) трансгенов, введенных с помощью смеси трансформантов A. tumefaciens. Такие анализы листьев были идеальными для быстрого исследования комбинаций генов и, как правило, позволяли прогнозировать метаболические пути, по большому счету разработанные для экспрессии в стабильно трансформированных растениях. Обычно каждая зона инфильтрирования была всего 2-3 см в диаметре, что давало общую сырую массу, составляющую от 50 до 100 мг на зону инфильтрирования. Небольшие количества свежего материала, такого как эти, не подходили для традиционных препаратов митохондрий растения, поскольку многочисленные стадии приводили к значительной потере митохондрий. Поэтому изобретатели разработали порядок одностадийной очистки митохондрий растения менее чем за 10 минут, начиная от небольших образцов до образцов массой 50-100 мг.
Наружная мембрана митохондрий растения имеет различные механизмы импорта и экспорта белка. Метаксин представляет собой специфичный для растений белок с массой примерно 40 кДа, обнаруженный на наружной мембране митохондрий растения и, возможно, участвующий в распознавании белков до их импорта в митохондрии (Lister et al., 2004). Белок, по-видимому, специфично локализован в митохондриях. Структурно метаксин имеет единственную перекрывающую мембрану область, локализованную по направлению к С-концу белка, при этом N-конец белка, вероятно, локализован в цитоплазме растения. Слияние GFP с N-концом метаксина приводит к появлению в митохондриях растения флуоресцентного сигнала, локализованного на наружной мембране (Lister et al., 2004). Авторы изобретения полагали, что N-конец метаксина, если он действительно локализован в цитоплазме, вероятно, будет доступен для связывания антитела и может позволить использовать способ очистки на основе аффинной метки. Кроме того, считалось, что помещение эпитопа на N-конце репортерного полипептида, например, вариант GFP mTurquoise, поможет протолкнуть эпитоп в цитоплазму. Метку TwinStrep выбирали в качестве метки для добавления к N-концу. Взаимодействие стрепавидина с меткой TwinStrep обеспечивало специфичное и прочное, но обратимое связывание с приложениями, описанными в способах очистки белков на основе аффинности (Schmidt et al., 2013; Schmidt и Skerra, 2007). Метка Twin-Strep в качестве трансляционного слияния обеспечивает прочное, но обратимое связывание со сконструированным связывающим субстратом StrepTactinXT, хотя он также может связываться со стрептавидином.
Авторы изобретения разработали слитой полипептид с несколькими компонентами, схематически показанными на Рисунке 15. Была разработана и изготовлена генетическая конструкция, кодирующая этот слитой полипептид. Сочетание синтеза гена и методов сборки Golden Gate было использовано для создания генетической конструкции, имеющей промотор 35S для экспрессии в растительных клетках и кодирующей слитой полипептид с метаксином последовательности распознавания TwinStrep-mTurquoise-TEV (конструкция SN197, SEQ ID NO:121). Метка Twin-Strep N-концевого эпитопа была включена, чтобы обеспечить опосредованную антителами аффинную очистку, а компонент mTurquoise позволял отслеживать очистку с помощью конфокальной микроскопии, а также продолжил N-конец метаксина дальше в цитозоль растения, а последовательность TEV распознавания протеазы позволила in vitro опосредованно расщеплять протеазой TEV полипептид, чтобы высвобождать митохондрии растений от магнитных микроносителей. Поскольку метаксин дикого типа внедряли в наружную мембрану митохондрий растения, то считалось, что экспрессия гена из конструкции SN197 в растительных клетках делает возможным очистку этой органеллы при условии, что слитой белок будет локализован на наружной мембране митохондрий. Это было неизвестно до тех пор, пока не было проведено исследование, как описано ниже.
Клетки A. tumefaciens, содержащие SN197, были инфильтрированы в листья N. benthamiana вместе как часть смеси клеток, содержащих отдельные конструкции для экспрессии супрессора сайленсинга p19, MTP-FAγ::GFP (конструкция pRA01) и локализованного в цитоплазме NifU::HA (SN211), каждый с OD, составляющим 0,1 и, следовательно, с общим OD, составляющим 0,4. Соответствующие контрольные смеси, содержащие некоторые, но не все компоненты, каждый с p19, были также инфильтрированы. Через четыре дня зоны инфильтрирования вырезали, получая образцы сырой массой примерно в 100 мг в кусочке листа размером 4 см х 2 см. Следующие стадии выполняли при 4 °C. Листовой материал растирали вручную в ступке пестиком, используя 500 мкл буфера KPBS. Этот буфер содержал 5,07 г KCl и 0,68 г KH2PO4 в 500 мл деионизированной воды, а pH был доведен до 7,25 с помощью 1 ммоль/л КОН. Суспензию центрифугировали на низкой скорости, 1000 g в течение 5 минут, чтобы осадить остатки клеточной стенки, но оставить большую часть митохондрий в суспензии. Надосадочную жидкость объемом 300 мкл добавляли к 50 мкл взвеси из магнитных микроносителей, покрытых стрептавидином (диаметр 2,8 мкм, магнитные микроносители с гладким покрытием, DynalBeads MyOne C1, код продукта 65002), в пробирку Eppendorf объемом 1,5 мл, а затем магнитные микроносители промывали один раз буфером KPBS. В установленное время магнитные микроносители в смеси собирали на стенке пробирки с помощью магнита, а остаток жидкости осторожно удаляли. Затем магнитные микроносители дважды промывали 1 мл KPBS, каждый раз собирая их, как и прежде, с помощью магнита, и, наконец, повторно суспендировали в 50 мкл KPBS. В качестве контрольного образца тот же порядок очистки магнитных микроносителей применяли к экстрактам листьев N. benthamiana, экспрессирующим pRA01 (кодирующий MTP-FAγ77::GFP), SN211 (кодирующая NifU::HA в цитоплазме) и p19, но без SN197.
Было проведено несколько экспериментов для определения оптимальных условий очистки митохондрий с использованием магнитных микроносителей. Во-первых, сравнивали различные магнитные микроносители, связывающие продукты TwinStrep. Было отмечено, что магнитные микроносители MyOne C1 превосходят Dynalbeads MyWay T1, M-280 и M-270, а также продукт IBT StreptaxtinXT-Agarose. Зависимость от времени связывания образца из SN197 с C1-магнитными микроносителями определяли с использованием инкубации в течение 1, 5, 10, 30 или 60 минут, и обнаружили, что максимальное и насыщенное связывание произошло через 5 минут. Сигнал GFP не был обнаружен после порядка очистки для образцов, в которых SN197 была исключена из смесей для инфильтрирования, что указывает на отсутствие неспецифичного связывания митохондрий с магнитными микроносителями. Конфокальная микроскопия показала, что флуоресцентные сигналы от mTurquoise (SN197) и GFP (MTP-FAγ77::GFP) были наибольшими при инкубации с магнитными микроносителями MyOne C1. С экстрактами инкубировали магнитные микроносители С1 различных концентраций. Выделение TwinStrep::mTurquoise::TEV::Метаксин и MTP-FAγ::GFP зависело от концентрации магнитного микроносителя, т. е. насыщения 50 мкл суспензии магнитным микроносителем MyOne C1, и поэтому впоследствии использовали это количество.
Стадии процесса очистки анализировали с помощью конфокальной микроскопии для оценки наличия полипептидов GFP и mTurquoise и собственной флуоресценции из хлоропластов растения. GFP и mTurquoise были обнаружены при длинах волн возбуждения 488 нм и 434 нм, соответственно. Образцы, полученные от инфильтрирования комбинацией конструкций SN197, pRA01 и p19, при измельчении с буфером KPBS и подвергнутых низкоскоростному центрифугированию, обогащали GFP-флуоресцирующими митохондриями. Наблюдалось только несколько интактных субклеточных органелл кроме митохондрий, например, хлоропласты и ядра, и несколько фрагментов продуктов распада клеток. После промывки магнитных микроносителей 2 мл буфера KPBS и оттягивания магнитом, конфокальная микроскопия полученной суспензии показала, что флуоресцирующие митохондрии физически прикреплены к магнитным микроносителям. После этой стадии очистки другие органеллы и продукты распада клеток не наблюдались.
Процесс очистки также подвергали анализу вестерн-блот. Для этого полипептиды, связанные с магнитными микроносителями, высвобождали и денатурировали в результате добавления 100 мкл буфера Лэммли (Пример 1) и нагрева образцов при 95 °C. Образцы растительного экстракта после измельчения, но до очистки, помеченные как «входной образец», также подвергали анализу вестерн-блот с использованием связывания антител с GFP для обнаружения полипептидов MTP-FAγ::GFP и mTurquoise:метаксин и анти-HA для обнаружения полипептида NifU::HA. Анализ вестерн-блот показал, что полипептид TwinStrep::mTurquiose::TEV::Метаксин легко обнаруживался при молекулярной массе примерно 80 кДа, что соответствует одному интактному трансляционному слитому белку. Полосу массой примерно 30 кДа наблюдали с антителом к GFP для образцов, включающих pRA01, что согласуется с ожидаемым размером митохондриально нацеленного MTP-FAγ::GFP. Кроме того, полосу массой примерно 42 кДа наблюдали с антителом HA из экстрактов, содержащих SN211, что соответствует ожидаемому размеру полипептида NifU::HA. Чтобы проверить наличие немитохондриальных белков как потенциальных загрязнителей, содержание цитоплазматического белка α-тубулина оценивали с помощью соответствующего антитела (Sigma Catalog No. T6074, моноклональное антитело клона B-5-1-2). Специфичная полоса для этого белка массой примерной 52 кДа наблюдалась только на дорожках, содержащих входной образец, а в очищенных экстрактах митохондрий не было обнаружено сигнала α-тубулина, что свидетельствует об очень хорошей очистке. Был сделан вывод, что использование слитого полипептида с метаксином, например, полипептида, кодированного SN197, позволило эффективно и быстро в небольших масштабах выделить и очистить митохондрии растения. Также был сделан вывод о том, что слитой полипептид способен встраиваться в наружную мембрану митохондрий растения после экспрессии генетической конструкции в растительных клетках и что N-концевая метка эпитопа TwinStrep доступна для покрытых стрептавидином магнитных микроносителей.
Когда выделенные и очищенные митохондрии были проанализированы с помощью протеомики, то оказалось, что образцы были сильно обогащены митохондриальными белками с очень низкими уровнями малой субъединицы рибулозо-1,5-бифосфаткарбоксилазы/оксигеназы. Это еще раз подтвердило высокую степень обогащения при использовании этого способа.
Пример 14. Ассоциация полипептидов NifS и NifU в митохондриях растительных клеток
Компоненты нитрогеназы содержат несколько металлокластеров, которые необходимы для функционирования. Белок нитрогеназы для фермента на основе молибдена, который осуществляет катализ, также известный как молибден-железосодержащий белок, представляет собой α2β2-тетрамер полипептидов NifD и NifK. В активном состоянии каталитический тетрамер содержит комплекс [Fe8S7], называемый P-кластером, на границе каждой α/β-субъединицы, а также FeMo-кофактор (FeMo-ко) в каждой α-субъединице. Компонент редуктазы нитрогеназы, также известный как железосодержащий белок, представляет собой гомодимер полипептидов NifH, который содержит субъединично-мостиковый кластер [Fe4S4]. Эти Fe-S и P-кластеры, а также FeMo-ко необходимы для переноса электронов для восстановления N2. Синтез и структура нитрогеназы рассмотрены в работе Rubio и Ludden (2005).
Правильная сборка и созревание этих металлокластеров представляет собой сложный процесс, в котором задействованы несколько дополнительных белков (Rubio и Ludden, 2008). Первым этапом процесса созревания является образование основных кластеров Fe-S. Это катализируется NifS и NifU. Эти два белка необходимы у бактерий для полной активности нитрогеназы. Затем кластеры Fe-S переносятся в NifH, NifB и, возможно, в NifD-NifK. NifS и NifU участвуют не только в сборке Mo-зависимой нитрогеназы, но также и в сборке нитрогеназы VFe и FeFe для синтеза их металлокластеров Fe-S (Kennedy и Dean, 1992).
Эти действия хорошо изучены у бактерий. NifS представляет собой пиридоксальфосфат (PLP, витамин B6), зависимую цистеин-десульфуразу, которая генерирует неорганический сульфид, необходимый для синтеза кластера Fe-S из цистеина. В результате реакции образуется аланин, побочный продукт реакции. Сульфид затем поставляется в NifU для последовательного образования кластеров [Fe2S2] и [Fe4S4]. Фермент NifS действует в бактериях как гомодимер.
NifU обеспечивает каркас для образования кластера [Fe4S4], функционирующего как гомодимер в бактерии. Его С-концевой домен может связывать один кластер [Fe2S2] на мономер. Кластеры [Fe2S2]в мономерах могут быть слиты с восстановлением и с образованием одного кластера [Fe4S4] на димер NifU. С-концевой домен NifU может содержать один кластер [Fe4S4] на мономер. Затем NifU передает кластеры [Fe4S4] в NifB для процессинга в ядре 8Fe на NifB, которое впоследствии используется для синтеза FeMo-ко. В дивергентном пути для кластеров Fe-S, один кластер [Fe4S4], связанный с N-концевыми или с C-концевыми каркасными доменами NifU, переносится в апо-NifH для созревания редуктазы нитрогеназы белка NifH (Smith et al., 2005). Было высказано предположение, что NifU также передает два кластера [Fe4S4] в NifD-NifK и что NifH конденсирует эту пару кластеров в зрелый P-кластер [Fe8-S7] (Dos Santos et al., 2004).
Сообщалось, что NifS и NifU образуют транзиентный комплекс у бактерий, но не компактный комплекс (Yuvaniyama et al., 2000). NifU не очищался совместно с NifS, когда NifS очищали от необработанных экстрактов, полученных из A. vinelandii (Dos Santos et al., 2012). Кроме того, специфичная иммунопреципитация NifU или NifS не приводила к копреципитации другого полипептида. Однако, когда выделенные и очищенные NifU и NifS объединяли in vitro и смесь подвергали эксклюзионной хроматографии, то обнаруживали гетеротетрамерный комплекс. Однако в этом эксперименте использовали очищенные белки. Отсутствовали сообщения о совместной экспрессии NifS и NifU в растительных клетках и о том, что они связываются друг с другом, а NifS ранее не очищался совместно с NifU от необработанных экстрактов.
Как описано в Примерах 2-4, нацеленный на митохондрии слитой полипептид NifU эффективно и достоверно процессировали с помощью MPP, а слитой полипептид NifS процессировали частично при продуцировании из генетических конструкций SN32 и SN31, соответственно. Слитые полипептиды имели MTP-FAγ51 для нацеливания на митохондрии и С-концевой эпитоп НА для обнаружения анализом вестерн-блот. В одном эксперименте, по меньшей мере, 90% процессированного полипептида NifU накапливалось в растворимой форме в митохондриях растения, хотя эта доля немного менялась от эксперимента к эксперименту, и некоторая часть (<50%) процессированного полипептида NifS накапливалась в растворимой форме. (Рисунок 3). Более того, было продемонстрировано, что полипептиды NifS и NifU, которые сохраняли мотив FAγ-scar9 на N-конце, являются функциональными в E. coli для поддержки активности нитрогеназы, и поэтому и NifS и NifU оставались активными с N-концевым продолжением из 9 аминокислот (Пример 4).
Основываясь на этих успехах, изобретатели разработали и провели следующим образом дальнейшие эксперименты для исследования продуцирования, процессинга, растворимости и функции NifS и NifU при введении в митохондрии в растительных клетках.
Конструирование плазмид, кодирующих слитые полипептиды с эпитопами TwinStrep
Были сконструированы и изготовлены две генетические конструкции для экспрессии кодируемых слитых полипептидов в растительных клетках с нацеливанием на митохондрии, одна кодирует слитой полипептид MTP-FAγ51::NifU::TwinStrep (SEQ ID NO:160), а другая слитой полипептид MTP-FAγ51::NifS::TwinStrep (SEQ ID NO:161). Аминокислотные последовательности из областей NifS и NifU слитых полипептидов были основаны на аминокислотных последовательностях белков Klebsiella oxytoca. Эпитоп TwinStrep (или метка) сокращенно обозначается в данном документе как «TS». Эпитоп TwinStrep выбирали, поскольку он обладает высокой связывающей аффинностью со смолой StrepTactinXT в основном при физиологических условиях и, таким образом, идеально подходит для очистки белков, содержащих эпитоп даже в низких концентрациях. Кроме того, условия элюирования были щадящими, что позволяло очищать белковые комплексы. Нуклеотидные последовательности кодирующих белок областей, были кодон-оптимизированы для улучшения экспрессии в растительных клетках. Каждая генетическая конструкция содержала промоторную последовательность 35S CaMV (Учетный № EC51288) для экспрессии в растительных клетках и область, кодирующую 51 аминокислоту MTP-FAγ51, слитую на 5’ с кодирующей областью Nif. Эти конструкции создались методами сборки Golden Gate (Weber et al., 2011; Engler et al., 2014) и аналогичными методами описанными выше. Эти конструкции были обозначены как SN166 для NifU и SN231 для NifS.
Была изготовлена другая конструкция (SN167), которая была аналогична SN166, за исключением того, что область MTP-FAγ51 была подвергнута мутации таким образом, что кодированный слитой полипептид имел замены аланина в последовательности MTP, которые не позволяли процессировать MPP в митохондриях, а мутировавшая область была обозначена как mFAγ51.
Продуцирование слитых полипептидов в растительных клетках, их процессирование и растворимость
Эти генетические конструкции вместе с другими исследовали на продуцирование и процессинг кодированных полипептидов в растительных клетках и на их растворимость. Как описано в Примерах 2 и 3, конструкция SN31, кодирующая слитой полипептид MTP-FAγ51::NifS::HA, была инфильтрирована в листья N. benthamiana, а белковые экстракты подвергали анализу вестерн-блот с антителом анти-НА. На мембранах для блоттинга наблюдали две полипептидные полосы. Они соответствовали по размеру непроцессированным и MPP-процессированным полипептидам (Пример 3). Процессированные и непроцессированные полипептиды NifS присутствовали как в растворимой, так и в нерастворимой белковой фракциях, что указывает на частичную растворимость. Напротив, когда SN166 вводили отдельно в листья N. benthamiana, слитой полипептид MTP-FAγ51::NifU::TS эффективно процессировался с помощью MPP, а полученный полипептид scar9-NifU::TS был почти полностью растворим, при этом scar9 включал линкер Gly-Gly, обусловленный использованным порядком клонирования. Как описано в Примере 4, полипептиды NifS и NifU, имеющие N-концевое продолжение из 9 аминокислот, были активны в обеспечении функции нитрогеназы для E. coli в сочетании с белками K. oxytoca дикого типа для других Nif. Также было показано, что метка His на С-конце NifS у A. vinelandii не мешает диазотрофному росту и сборке кластеров FeS на NifH (Smith et al., 2005).
Генетические конструкции SN166 и SN167 вводили отдельно в листья N. benthamiana, чтобы подтвердить эффективность последовательности MTP в полипептиде, кодированном SN166, и влияние митохондриального нацеливания на растворимость и очистку в колонке StrepTactinXT. Белки экстрагировали из тканей листа в неденатурирующих условиях. Буфер для экстракции содержал 100 ммоль Трис-HCl pH 8,0, 150 ммоль NaCl, 5% (об./об.) глицерина, 2 ммоль TCEP, 1% (мас./об.) PVP (средняя молекулярная масса 40 кДа) и 0,1% Tween® 20. Колонку StrepTactinXT емкостью 2 мл промывали буфером, содержащим 100 ммоль Трис pH 8,0, 150 ммоль NaCl и 2 ммоль TCEP (промывочный буфер), а затем загружали экстрактом белка из SN166 или, отдельно, из SN167. После промывки колонки для удаления несвязанных белков, связанные белки элюировали промывочным буфером, содержащим 50 ммоль биотина. Образцы, содержащие белок, концентрировали до объема 200-500 мкл, используя концентратор Amicon Ultra 10 kD MWCO емкостью 4 мл. Аликвоты по 20 мкл подвергали электрофорезу SDS-PAG и анализу вестерн-блот с использованием антитела Streptactin HRP. Двойные гели окрашивали кумасси синим для окрашивания белков.
Анализ вестерн-блот (Рисунок 16, верхняя панель) показал, что слитой полипептид NifU::TwinStrep действительно был очищен от тканей, инфильтрированных из SN166, с использованием колонки StrepTactinXT. Экстракт из инфильтрированных тканей SN167 давал небольшое содержание очищенного белка NifU::TwinStrep, который, по-видимому, имел в основном непроцессированную форму. Соответствующие гели, окрашенные кумасси синим (Рисунок 16, нижняя панель), подтвердили, что в процессе очистки произошла высокая степень обогащения.
Срезы геля вырезали из геля, окрашенного кумасси, и полипептиды в этих срезах подвергали анализу на наличие N-концевой аминокислоты. Это подтвердило, что слитой полипептид MTP-FAγ51::NifU::TwinStrep, кодированный SN166, был расщеплен MPP в намеченном сайте в последовательности MTP, поскольку очищенный полипептид имел N-концевую последовательность, полученную в результате предполагаемого процессинга.
На основании этих данных был сделан вывод о том, что слитые полипептиды NifS и NifU, которые были нацелены на митохондрии, действительно экспрессировались в растительных клетках и процессировались в митохондриях, и находились в достаточно растворимой форме для очистки.
Совместная экспрессия MTP::NifU::TwinStrep и MTP::NifS::HA в растительных клетках - NifS и NifU ассоциированы в митохондриях растения
Для оценки экспрессии, процессинга, растворимости и стабильности, а также исследования возможной ассоциации слитых полипептидов NifS и NifU при их совместном продуцировании в митохондриях растения, генетическую конструкцию SN31 (Пример 2), кодирующую полипептид MTP-FAγ51::NifS::HA и SN166, кодирующую полипептид MTP-FAγ51::NifU::TS, совместно инфильтрировали в листья N. benthamiana, используя способ, описанный в Примере 1. Белковые экстракты из листьев были приготовлены и исследованы на наличие комплексов NifS-NifU, прежде всего, путем проведения аффинной очистки NifU с использованием колонки StrepTactinXT, а затем исследования совместной очистки полипептида NifS с использованием способа, описанного в Примере 1. Вкратце, в первом эксперименте 12 г сырой массы листа процессировали в анаэробных условиях с использованием неденатурирующего буфера для экстракции. Вторую, повторную очистку начинали с 16,6 г сырой массы листа. Третью очистку проводили с использованием 23 г сырой массы листа, при этом используемый буфер немного отличался тем, что добавляли Fe2+ и L-цистеин в количестве 2 ммоль и 0,5 ммоль, соответственно. В каждом эксперименте отфильтрованный лизат пропускали через колонку StreptactinXT (IBA Lifesciences) для удержания полипептида NifU его эпитопом TS. После промывки колонки связанные белки элюировали буфером, содержащим биотин, а затем концентрировали, как описано выше. Образцы сохраняли на каждой стадии процесса очистки, в частности образцы: (i) общего экстрагируемого белка в начале эксперимента, (ii) осажденные продукты распада клеток после первого центрифугирования, (iii) раствор входящего белка, фракция которого была растворима в буфере для экстракции до прохождения через колонку, (iv ) проточная фракция, которая не связывалась с колонкой, и (v) концентрированный элюат после элюирования биотином. Образцы обрабатывали SDS и нагревали до 95°C перед электрофорезом SDS-PAG и анализом вестерн-блот.
Очищенный и концентрированный образец NifU после третьей очистки имел некоторый видимый коричневый цвет, что указывало на присутствие кластеров Fe-S.
Дополнительные аликвоты этих образцов подвергали анализу вестерн-блот с иммунологическим анализом, используя антитело анти-Strep или антитело анти-HA. Анализы вестерн-блот из первого и третьего экспериментов по очистке показаны на Рисунках 17 и 18. Третий эксперимент по очистке проводили в буфере для экстракции в присутствии 0,5 ммоль L-цистеина и 2 ммоль Fe2+. Анализ вестерн-блот показал, что оба белка присутствовали в растворимой фракции после экстракции из листа. Процессированная и непроцессированная формы присутствовали в растворимой фракции для NifS, в то время как для NifU присутствовала только процессированная форма, что указывает на эффективность процессинга. Антитело анти-Strep обнаружило полипептид scar9-NifU-TwinStrep в неочищенных образцах, а также в образце, элюированном из колонки. Интенсивность сигнала от элюата была очень высокой, что указывало на то, что полипептид scar9-NifU-TwinStrep был очищен и сконцентрирован из растительных экстрактов. Подвижность полипептида в гелях при электрофорезе соответствовала процессингу митохондрий в последовательности MTP, а процессинг оказался почти полным.
После воздействия на мембрану антителом анти-НА, которое было примерно в 20 раз более чувствительным, чем антитело анти-Strep, был обнаружен полипептид с меткой НА, размер которого соответствовал митохондриальному процессингу полипептида NifS, т. е. scar9-NifS::HA. В образце не было обнаружено непроцессированной формы слитого полипептида NifS. Поскольку полипептид NifS, использованный в этом эксперименте, не содержал метки Strep, то эти результаты показали, что NifS и NifU образовали комплекс и что полипептид NifS очищался совместно с NifU. Важно отметить, что процессированная форма, scar9-NifS::HA, была значительно обогащена в элюате из колонки по сравнению с непроцессированной формой исходя из соотношения двух форм во входном образце до очистки в колонке. Эти наблюдения были неожиданными для изобретателей исходя из сообщений о бактериях, экспрессирующих NifS и NifU, которые не продемонстрировали ассоциации полипептидов. Они пришли к выводу, что в анаэробных неденатурирующих условиях, использованных в эксперименте по экстракции белка: (i) слитой полипептид NifS очищали совместно с полипептидом scar9-NifU::TS, что указывает на ассоциацию двух полипептидов при совместной экспрессии в растительных клетках с митохондриальным нацеливанием, (ii) MPP-процессированная форма полипептид NifS, scar9-NifS::HA представляла собой форму, которая ассоциировалась с полипептидом NifU, и (iii) процессированные полипептиды NifU и процессированные полипептиды NifS продуцировались, по меньшей мере, частично в растворимой форме в митохондриях для обеспечения наблюдаемой ассоциации. Для наблюдения (ii) существовало, по меньшей мере, три возможных объяснения. Во-первых, непроцессированный MTP-FAγ51::NifS мог быть не в состоянии взаимодействовать с NifU из-за стерического несоответствия или нарушенной укладки. Во-вторых, непроцессированная форма могла не быть импортирована в митохондрии, где был локализован полипептид NifU, и, в-третьих, непроцессированная форма NifS могла быть недостаточно растворимой и, следовательно, не могла взаимодействовать с NifU, или любая комбинация этих причин.
Авторы изобретения не знали о каких-либо предыдущих сообщениях о выделении комплекса NifS-NifU из митохондрий растения или даже из любой клетки.
Образцы после первой очистки снова подвергали электрофорезу SDS-PAGE в денатурирующем геле. На этот раз гели окрашивали кумасси синим (Рисунок 18, панель C), а области геля, соответствующие процессированным полипептидам NifU и NifS, анализировали с помощью протеомики для идентификации как введенных полипептидов, так и любых эндогенных белков, которые совместно очищали в колонке. Срезы геля обрабатывали, как описано в Примере 1, включая расщепление трипсином, и анализировали с помощью LC-MS/MS. Анализ выявил присутствие пептида ISTQVVR (SEQ ID NO:119), предсказанного при триптическом расщеплении пептида scar (SEQ ID NO: 42) на N-конце, показывая, что NifS и NifU процессировались точно в предсказанном сайте расщепления MPP в MTP. Нацеленная MRM подтвердила идентичность триптических пептидов и тем самым подтвердила присутствие расщепленных полипептидов в областях, ожидаемых при электрофорезе SDS-PAGE.
Эксклюзионная хроматография по размеру
Чтобы еще раз подтвердить, что белковый комплекс образовывался между NifS и NifU, образец концентрированного элюата подвергали эксклюзионной хроматографии по размеру с высоким разрешением, используя в качестве колонки Superdex 200 Increase 3.2/300. Калибровку колонки проводили с помощью маркеров размера нативного белка (Biorad Gel Filtration Standard Cat. № 151-1901). Образцы фракций из колонки дополнительно анализировали электрофорезом SDS-PAGE в денатурирующем геле. Хроматограмма и анализ вестерн-блот показали, что NifS и NifU образовывали комплекс, поскольку белок NifS элюировался с более высокой молекулярной массой, чем ожидалось для NifS. Это указывает на то, что гетеротетрамер образовывался ассоциацией 2-х полипептидов NifS и 2-х полипептидов NifU.
Спектроскопия в УФ- и видимой области обнаружила железо-серные кластеры на NifU
Элюат, содержащий NifU и NifS, очищенные в колонке StreptactinXT, из четвертого эксперимента, переносили в колонку PD10 (GE Healthcare), уравновешенную 50 ммоль Tris-HCl pH 8,0 и 300 ммоль NaCl для удаления биотина, избытка Fe2+ и цистеина. Спектр получали с использованием анаэробной кюветы с завинчивающейся крышкой и перегородкой с длиной 1 см на спектрофотометре УФ и видимой областей спектра Cary 100 Bio. Спектр показал один основной пик при длине волны 280 нм, как и ожидалось для белков из-за поглощения триптофаном, фенилаланином и цистеином. Кроме того, второй пик наблюдался при длине волны 325 нм и «плечо» при 420 нм и 460 нм, что указывало на присутствие кластеров Fe-S в NifU.
Дальнейшие исследования на ассоциацию полипептидов NifS и NifU путем очистки прежде всего NifS
Как описано выше, была сконструирована и изготовлена генетическая конструкция, обозначенная SN231, которая кодировала слитой полипептид MTP-FAγ51::NifS::TS для транзиентной экспрессии в растительных клетках. Эта конструкция была аналогична SN166, которая кодировала слитой полипептид MTP-FAγ51::NifU::TS, за исключением того, что она имела последовательность NifS, а не последовательность NifU. SN231 и SN32 совместно инфильтрировали в листья N. benthamiana как для описанной выше комбинации SN31/SN166, а белковые экстракты получали описанным выше способом. Надосадочную жидкость пропускали через колонку StrepTactinXT для очистки слитого полипептида NifS, содержащего эпитоп TwinStrep. Образцы элюированных и концентрированных белков подвергали анализу вестерн-блот и исследовали антителами анти-Strep и анти-HA. Мембрана для блоттинга (Рисунок 19) показала присутствие процессированного полипептида scar9::NifU::HA в элюате, а также полипептида scar9::NifS::TS, что снова указывает на ассоциацию процессированных полипептидов NifS и NifU в экстрактах из растительных клеток.
Элюат после этой очистки также подвергали эксклюзионной хроматографии по размеру, как описано выше, а фракции подвергали анализу вестерн-блот с использованием антитела анти-Strep и антитела анти-НА. Анализ вестерн-блот подтвердил, что NifU и NifS образовали комплекс.
В будущем очищенные полипептиды NifS и NifU будут анализироваться способом масс-спектрометрии с индуктивно связанной плазмой (ICP-MS) для определения содержания железа в белке и способом гамма-резонансной спектроскопии для подтверждения наличия, типа и окислительно-восстановительного статуса Fe- S-кластеров, связанных с полипептидами.
Образование кластера можно показать в реакциях in vitro с добавлением Fe2+ и L-цистеина. В одном эксперименте полипептид NifH дикого типа очищают от A. vinelandii, а кластеры Fe-S удаляют хелатированием с получением полипептида апо-NifH. Комплекс NifD-NifK дикого типа также очищают от A. vinelandii. Анализы ARA in vitro показывают, что очищенный полипептид NifU, очищенный от клеток N. benthamiana, как описано выше, способен передавать кластеры Fe-S полипептиду апо-NifH, тем самым восстанавливая активность NifH в качестве редуктазы нитрогеназы для активности ARA.
Пример 15. Продуцирование гомоцитрата путем экспрессии NifV в растительных клетках
Введение
Упоминаемая здесь (R)-2-гидрокси-1,2,4-бутан-трикарбоновая кислота, известная как гомоцитрат, требуется для активности всех известных нитрогеназ, а именно молибденовой (Mo-Fe), ванадиевой (V -Fe) и железистой нитрогеназы (Fe-Fe), соответственно (Hu и Ribbe, 2016). Белок нитрогеназы для фермента на основе Mo, который выполняет восстановление азота, представляет собой α2β2-тетрамер полипептидов NifD и NifK, который содержит FeMo-кофактор (FeMo-ко) в каждой α-субъединице, а также комплекс [Fe8S7], называемый P-кластер на границе каждой α/β-субъединицы. FeMo-ко, содержащий молекулу гомоцитрата, необходим для восстановления N2.
Гомоцитрат (HC) формирует часть основных кофакторов нитрогеназы FeMo-ко, FeV-ко и FeFe-ко в экспрессирующих нитрогеназу бактериях, связываясь с атомами Mo, V или Fe кофактора через его 2-гидрокси- и 2-карбоксильные группы. FeMo-ко, FeV-ко и FeFe-ко находятся на сайтах катализа, и считается, что эти три кофактора связывают, активируют и восстанавливают N2 в основном одинаковым образом. FeMo-ко, также известный как M-кластер Mo-нитрогеназы, содержит подкластеры [Fe4S3] и [MoFe3S3], соединенные тремя мостиковыми атомами неорганического сульфида, называемыми «опоясывающими сульфидами», и одним межузловым атомом карбида (Hu и Ribbe, 2016) с образованием кофактора, имеющего химическую формулу HC-Mo-Fe7-S9-C. Ванадиевая нитрогеназа, включая ее кофактор FeV-ко, недавно была кристаллизована (Sippel и Einsle, 2017; Sippel et al., 2018). FeV-ко имеет ядро металл-сера почти идентичное FeMo-ко, за исключением замены атома ванадия на атом молибдена и карбонат-иона вместо одного из опоясывающих сульфидов. Таким образом, FeV-ко представляет собой кластер [HC-V-Fe7-S8-CO3-C] с молекулой гомоцитрата, присоединенной к атому ванадия в результате лигандного взаимодействия. В случае полипептида VnfDAzotobacter vinelandii, который является частью каталитического фермента V-нитрогеназы (VnfDGK), гомоцитрат металлокластера координируется с аминокислотами C257 и H423 VnfD. Эти лигандные аминокислоты высоко консервативны по сравнению с NifD Mo-нитрогеназы. Mo- и V-нитрогеназы различаются по реакционной способности по отношению к монооксиду углерода (CO), который ингибирует первый, но преобразуется в углеводороды вторым (Sippel et al., 2018). Гомоцитрат аналогичным образом образует часть FeFe-ко, а кофактор связывается с полипептидом AnfD аналогичным образом. Fe-нитрогеназа имеет более низкую активность восстановления N2 по сравнению с V-нитрогеназой, которая, в свою очередь, менее активна, чем Mo-нитрогеназа, что позволяет предположить, что организмы, которые оснащены всеми тремя системами, прибегают к предпочтительной экспрессии, которая зависит от относительной биодоступности Mo, V и Fe. Например, бактерия A. vinelandii может экспрессировать каждую из нитрогеназ Mo-, V- и Fe-, но каждая из них при разной питательной среде, V-нитрогеназа только в условиях ограничения по молибдену, а Fe-нитрогеназа только в том случае, если ограничены и Mo и V.
У свободноживущих азотфиксирующих бактерий гомоцитрат продуцируется генным продуктом NifV, ферментом, который конденсирует ацетил-CoA и α-кетоглутарат (αKG) с образованием гомоцитрата (Zheng et al., 1997). NifV представляет собой единственный генный продукт, необходимый для синтеза гомоцитрата у этих бактерий. Активность гомоцитрат-синтазы можно измерить с помощью ферментных анализов, как описано в работе Zheng et al. (1997). Мутанты A. vinelandii nifV не способны продуцировать какую-либо форму полностью активной нитрогеназы, но активность всех трех нитрогеназ восстанавливается добавлением гомоцитрата в среду для выращивания (Zheng et al., 1997). В отсутствие добавленного гомоцитрата мутантные бактерии nifV проявляли аномальные реакции, опосредованные нитрогеназой, включая измененную специфичность субстрата и специфичность ингибитора. Мутантные бактерии восстанавливали ацетилен и выделяли H2, но не восстанавливали N2 (McLean и Dixon, 1981). Эти измененные активности были связаны с включением эндогенных молекул, связанных с гомоцитратом, например, цитратом в металлокластере (Hoover et al., 1988). Считается, что гомоцитрат уникален по своей способности правильно размещать субстрат N2 в активном сайте и поэтому необходим для полного и правильного функционирования нитрогеназ.
NifV A. vinelandii является наиболее изученным NifV (Zheng et al., 1997; SEQ ID NO:163) и называется здесь AvNifV. Сверхэкспрессия полипептида AvNifV в E. coli приводила к образованию димерного белка с молекулярной массой примерно 89 кДа, при этом мономер имел молекулярную массу 44 кДа. Фермент распадался под действием кислорода, теряя примерно 50% своей активности после двухчасового воздействия воздуха, содержащего 21% кислорода. Добавление MoO4-2, Fe2+ или Mg2+ в реакционную среду не влияло на эту чувствительность к кислороду из за его конденсационной активности. Кинетика реакции показала, что AvNifV имел Km 0,06 ммоль для ацетил-CoA и 2,24 ммоль для αKG. NifV может также конденсировать ацетил-CoA с другими кетокислотными субстратами, например, оксалоацетатом и α-кетоадипатом (Zheng et al., 1997).
В симбиозах бобовых и микоризы, например, среди Lotus japonicus и Mesorhizobium loti, бактериальный партнер не обладал активностью гомоцитрат-синтазы, кодированной геном NifV. Вместо этого растение-хозяин L. japonicus экспрессирует гомоцитрат-синтазу, LjFEN1, чтобы поставлять эту незаменимую органическую кислоту для фиксации азота микоризой в клубеньках (Hakoyama et al., 2009). Полипептид LjFEN1 довольно отдаленно родственен NifV A. vinelandii, эти два полипептида имеют примерно 36% аминокислотной идентичности. LjFEN1 имеет 540 аминокислотных остатков и молекулярную массу примерно 58,6 кДа. Последовательность сигнального пептида не была обнаружена в гене, кодирующем LjFEN1, что указывает на вероятность наличия цитозолевого белка. L. japonicus имеет два ортолога FEN1, а именно по Учетными №№ AK339695 и AK339656, которые на 81% и 71% идентичны аминокислотной последовательности LjFEN1, соответственно. Филогенетический анализ показал, что LjFEN1 произошел от AK339695. При симбиозе между растениями L. japonicus, мутировавшими в LjFEN1 и M. loti, продуцировались полностью функциональные клубеньки с обнаруживаемой активностью нитрогеназы, если меньший по величине партнер симбиоза нес гетерологичную копию гена AvNifV или гена FEN1.
В отличие от многих других эукариот, грибы, например, дрожжи Saccharomyces cerevisiae, продуцируют гомоцитрат в качестве промежуточного звена на пути биосинтеза лизина через NifV-подобный фермент (Thomas et al., 1966; Verhasselt et al., 1995). Дрожжевые мутанты в гене ORF D1298, кодирующем NifV-подобный фермент, который функционирует на этом пути, были дополнены сверхэкспрессией LjFEN1.
Геномный анализ многочисленных видов растений показывает, что только те растения, которые участвуют в симбиотических отношениях с бактериями, экспрессируют активную гомоцитрат-синтазу, например LjFEN1, и что NifV-подобные гены не обнаруживаются у небобовых растений (Hakoyama et al., 2009). Кроме того, у высших растений не было выявлено метаболических путей синтеза лизина через гомоцитрат в качестве промежуточного продукта. В соответствии с этими сообщениями, исследование геномной последовательности N. benthamiana (Naim et al., 2012) не выявило никаких гомологов NifV или FEN1. Ближайшим геном, идентифицированным с точки зрения гомологии, был ген (QUT N. benthamiana Genome и Transcriptome DB Учетный №. P72026), который был гомологичен гену, кодирующему фермент 2-изопропилмалат-синтазы (EC. 2.3.3.13), вовлеченному в биосинтез лейцина, но не в синтез гомоцитрата. Авторы изобретения пришли к выводу, что N. benthamiana обычно не продуцирует гомоцитрат с помощью NifV или FEN1-подобных ферментов. За исключением единственного отчета, относящегося к стручкам ванили (Palama et al., 2009), изобретатели не знают ни одного отчета о гомоцитрате, продуцированном не из бобовых, в том числе из отчетов о табаке, хлопке и зерновых. Нет известных сообщений об использовании FEN1 или NifV для продуцирования гомоцитрата у небобовых культур.
Результаты исследования
Как описано в Примерах 2-4 выше, слитой полипептид NifV на основе аминокислотной последовательности K. oxytoca (KoNifV; SEQ ID NO:13), нацеленный на митохондрии в растительных клетках, эффективно ( > 90%) и достоверно процессировали с помощью MPP при продуцировании из генетической конструкции SN142. Слитой полипептид, транслированный при экспрессии генетической конструкции, имел N-концевой MTP-FAγ51 для нацеливания на митохондрии и С-концевой эпитоп НА для обнаружения анализом вестерн-блот. Более того, было продемонстрировано, что полипептид NifV на основе аминокислотной последовательности K. oxytoca с мотивом FAγ-scar9, слитым на N-конце, является функциональным в E. coli для поддержания уровней активности нитрогеназы, близких к дикому типу, обеспечивая около 90% активности в системе MIT2.1 по сравнению с диким типом, и поэтому слитой полипептид NifV оставался активным с N-концевым продолжением из 9 аминокислот (Пример 4). Однако процессированный полипептид KoNifV накапливался в нерастворимой форме в митохондриях растения (Рисунок 3).
Нерастворимость слитого полипептида NifV K. oxytoca в клетках N. benthamiana рассматривалась авторами как проблема для введения в действие функции нитрогеназы в растительных клетках, поскольку практически нерастворимый полипептид вряд ли обеспечил достаточную ферментативную функцию для синтеза гомоцитрата. Поэтому авторы изобретения искали более растворимые полипептиды NifV с помощью экспрессии природного NifV и других HCS-подобных вариантов, слитых с теми же последовательностями эпитопа MTP и HA, для локализации и обнаружения в митохондриях.
Выбор вариантных последовательностей NifV
В базах данных последовательностей был проведен поиск вариантных последовательностей NifV и других ферментов гомоцитрат-синтазы (HCS), связанных с аминокислотной последовательностью KoNifV. Последовательности получали от самых разных бактерий и дрожжей, в том числе от термотолерантных бактерий. Последовательности полипептида NifV были извлечены из базы данных UniProt с использованием NifV в качестве запроса, а доступ к базе данных был получен 14.09.2018. NifV/HCS-подобные аминокислотные последовательности в количестве 2044 штук были идентифицированы и извлечены из базы данных. Для выбора и исследования репрезентативных последовательностей была создана белковая сеть на основе сходства белков, что привело к кластеризации NifVα-HCS-подобных полипептидов на основе сходства последовательностей. Для этого аминокислотные последовательности были выровнены с помощью ПО MAFFT (Программа множественного выравнивания аминокислотных или нуклеотидных последовательностей ), версия 7 с использованием сервера mafft.cbrc.jp/alignment/server/large.html?aug31. Использовали стратегию G-large-INS-1 для менее чем 10 000 последовательностей, короче чем 5 000 сайтов. Выходные данные были преобразованы из формата.pir в формат.phy с помощью онлайн-конвертера последовательностей (www.hiv.lanl.gov/content/sequence/ FORMAT_CONVERSION/form.html).
Программное обеспечение Cytoscape (https://cytoscape.org) использовали для визуализации кластеров последовательностей, родственных друг другу. Для вычисления матриц расстояний и подготовки данных во входных файлах для Cytoscape, была использована программа PHYLIP/protdist для вычисления матрицы расстояний Кимуры для последовательностей NifV. Выходной файл изменяли в блокноте Notepad, чтобы подготовить соответствующий входной формат для aMATReader в ПО Cytoscape. Затем матрицу расстояний изменяли в Excel для уменьшения размера файла и определения подгрупп: все значения, превышающие 0,1, были удалены, в результате чего были созданы подгруппы, а избыточные последовательности были удалены.
Репрезентативная HCS-подобная аминокислотная последовательность была выбрана из каждого из шести кластеров HCS и родственных последовательностей. Кроме того, были отобраны три HCS-подобные последовательности Methanocaldococcus infernus, поскольку они с большей вероятностью считались термотолерантными и, возможно, оставались стабильными и растворимыми, а также последовательности NifV из K. oxytoca (KoNifV) и A. vinelandii (AvNifV) для сравнения. Вариант KoNifV (Учетный №. WP_004138778; SEQ ID NO:164) также был идентифицирован на основе аминокислотной последовательности в конструкции MIT2.1 бактериальной экспрессии. Аминокислотные последовательности KoNifV в EC38020 и NifV в MIT2.1 различались по аминокислотам 155-157 и 232-236 относительно SEQ ID NO:13, но в остальном были идентичными. Была также выбрана последовательность Saccharomyces cerevisiae HCS (ScHCS), соответствующая гену Lys21p S. cerevisiae, обозначенному как D1298 в работе Verhasselt et al. (1995). Гомологичный фермент Lys20 в S. cerevisiae оказался более активным и менее негативно регулируется лизином.
Выбранные последовательности перечислены в Таблице 15 вместе с процентной идентичностью по отношению к KoNifV из EC38020 (SEQ ID NO:13). Выравнивание аминокислотных последовательностей показано на Рисунке 20, на котором представлены высококонсервативные аминокислоты. Очевидно, что выбранные последовательности охватывают широкий спектр NifV/HCS-подобных последовательностей.
Таблица 15. NifV и NifV/HCS-подобные последовательности, выбранные для исследования
Конструирование плазмид, кодирующих слитые полипептиды с NifV и NifV/HCS-подобными последовательностями
Слитые полипептиды, имеющие выбранные последовательности NifV и NifV/HCS, перечислены в Таблице 15, а последовательности, имеющие MTP-FAγ51 на каждом N-конце, затем исследовали на их способность экспрессироваться в растительных клетках, на их процессинг с помощью MPP в митохондриях и на их продуцирование гомоцитрата. Растворимость каждого полипептида, нацеленного на митохондрии, также исследовали с использованием способа, описанного в Примере 1. Эти эксперименты выполняли в результате создания генетических конструкций, кодирующих эти последовательности и экспрессирующих их в листьях N. benthamiana. Каждый кодированный слитой полипептид имел идентичный эпитоп HA для обнаружения с помощью антитела анти-HA, локализованного между MTP и NifV/HCS-подобной последовательностью, за исключением слитого полипептида KoNifV, кодированного конструкцией SN142, которая имела эпитоп HA на своем С-конце (Таблица 15). Таким образом, этот эксперимент был разработан для исследования, будут ли N- или C-концевые продолжения каждой NifV/HCS-подобной последовательности по-прежнему допускать продуцирование гомоцитрата в растительных клетках. Параллельный набор генетических конструкций (Таблица 15) был создан для экспрессии полипептидов нацеленных в цитоплазму, лишенных последовательности MTP-FAγ51 на N-концах, но содержащих N-концевой эпитоп НА. Таким образом, каждый слитой полипептид сравнивали по его экспрессии и функции с его соответствующим цитоплазматическим полипептидом, лишенным последовательности MTP.
Последовательность DNA для каждого слитого полипептида синтезировали с использованием кодон-оптимизации для экспрессии в растении, которая была совместима с методами сборки Golden Gate. Генетические конструкции создавали с использованием методов сборки Golden Gate. Компоненты DNA, за исключением SN142, располагались для сборки в порядке от 5’ до 3’: промотор 35S CaMV (EC51288); химерная последовательность, кодирующая эпитоп MTP-FAγ51 и HA, за которой следовал линкер GG (EC38095); кодон-оптимизированные кодирующие области для NifV/HCS-подобного варианта; и, наконец, области CaMV 3’ последовательности полиаденилирования/терминатора транскрипции (EC41414). Компоненты собирали в требуемые генетические конструкции и вставляли в векторы экспрессии с использованием рестрикционного клонирования типа IIS в соответствии с методами сборки Golden Gate (Weber et al., 2011). Полученные конструкции приведены в Таблице 16. Молекулярные массы кодированных слитых полипептидов до и после MPP-процессирования рассчитвали с использованием аппарат ExPASy compute pl/Mw (web.expasy.org/compute_pi/) с моноизотопной настройкой.
Таблица 16. Генетические конструкции для транзиентной экспрессии NifV/HCS-подобныхслитых полипептидов в листьях N. benthamiana или в стабильно трансформированных растениях. MTP, митохондриальный нацеливающий пептид FAγ51; Mw, молекулярная масса; нд, не применяется.
Экспрессия в листьях N. benthamiana и исследование на растворимость и продуцирование гомоцитрата
Каждую генетическую конструкцию вводили в листья N. benthamiana с помощью Agrobacterium, используя способы, описанные в Примере 1. Образцы листьев собирали через 5 дней после инфильтрирования, а экстракты белков получали и подвергали анализу вестерн-блот с использованием антител анти-НА (Рисунки 21 и 22). Параллельные образцы листьев собирали для экстракции метаболитов и измерения уровней гомоцитрата способом GC-MS/MS, как описано ниже.
Все исследованные слитые полипептиды легко обнаруживались с помощью анализа вестерн-блот и, следовательно, эффективно экспрессировались в растительных клетках как для митохондриально-нацеленных, так и для нацеленных в цитоплазму полипептидов. Как наблюдалось ранее (Пример 3), митохондриально-нацеленный слитой полипептид NifV K. Oxytoca, продуцировали на хороших уровнях и эффективно процессировали MPP, но он был практически нерастворим в растительных клетках. Сходным образом митохондриально нацеленные слитые полипептиды MiHCS2, MiHCS3 и MaHCS также экспрессировались на хорошем уровне и, по-видимому, подвергались процессингу, но не растворились. Слитые полипептиды NsHCS и MiHCS1, по-видимому, процессировались, но растворялись лишь частично. Напротив, митохондриально нацеленные слитые полипептиды TbHCS, TpHCS и CtHCS, по-видимому, процессировались и, по существу, растворялись. Митохондриально нацеленный HCS S. cerevisiae (ScHCS), по-видимому, экспрессировался на более низком уровне, чем другие полипептиды, но эффективно процессировался и растворялся. Слитой полипептид NifV Azotobacter vinelandii (AvNifV) экспрессировался на хорошем уровне, эффективно процессировался и был частично растворим (~ 50%) при нацеливании на митохондрии растения с использованием MTP-FAγ51. Аналогичным образом, HCS Chlorobaculum tepidum (CtHCS) хорошо экспрессировался, эффективно процессировался и растворялся при нацеливании на митохондрии растения с использованием MTP-FAγ51.
В отличие от митохондриально нацеленных полипептидов, большинство полипептидов, нацеленных на цитоплазму, были растворимы или, по меньшей мере, частично растворимы, включая полипептид KoNifV (Рисунок 22). Авторы изобретения пришли к выводу, что нерастворимость в некоторых случаях связана с локализацией митохондрий и что полипептиды могут демонстрировать разные уровни растворимости в двух локализациях. В целом, интенсивность сигналов полипептидов, нацеленных на цитоплазму, была ниже по сравнению с соответствующими митохондриально нацеленными полипептидами. Исключение составили ScHCS, MiHCS1 и KoNifV, где полипептиды, нацеленные на цитоплазму, обладали лучшими уровнями экспрессии по сравнению с митохондриально нацеленными аналогами.
Газовая хроматография с тандемной масс-спектрометрией (GC-MS/MS) для измерения уровней гомоцитрата
Для измерения уровней гомоцитрата в образцах листьев после введения гена и, таким образом, демонстрации активности HCS в отношении митохондриально-нацеленных слитых полипептидов или слитых полипептидов нацеленных на цитоплазму был разработан и утвержден способ GC-MS/MS. Полярные метаболиты, включая любой гомоцитрат, экстрагировали по 10 объемов на сырую массу листьев (об./мас.) экстракционного раствора, которые содержали в смеси метанол:H2O (1:1 об./об.): 22 мкмоль/л лимонной кислоты D4 (Cambridge Isotope Laboratories Inc., Учетный №. DLM-3487), 36 мкмоль/л 13C фумаровой кислоты (Cambridge Isotope Laboratories Inc., Учетный №. CLM-1529), 23 мкмоль/л 13C сорбита (Cambridge Isotope Laboratories Inc., Учетный №. CLM-1565), 31 мкмоль/л D3 аспарагиновой кислоты (Cambridge Isotope Laboratories Inc., Учетный №. DLM-832) и 54 мкмоль/л D5 глицина (Cambridge Isotope Laboratories Inc., Учетный №. DLM. -280) в качестве внутренних стандартов. Образцы листьев гомогенизировали с экстракционным раствором в микроцентрифужных пробирках объемом 1,5 мл с использованием гомогенизатора Qiagen tissue lyser и 3 мм магнитных микроносителей из карбида вольфрама. Образцы листьев гомогенизировали при 1/20 об/мин дважды в течение трех минут, меняя положения пробирок внутри штативов, которые были предварительно охлаждены до -80 °C. После гомогенизации образцы центрифугировали при 10000 × g в течение 30 минут при 4°C для удаления твердого вещества, а полученную надосадочную жидкость, содержащую метаболиты, собирали и хранили до анализа при -80 °C. Тридцать мкл каждого образца надосадочной жидкости сушили в вакуумном концентраторе для дериватизации метаболитов, которую проводили вручную следующим образом. К каждому высушенному образцу добавляли 10 мкл 20 мг/мл метоксиамингидрохлорида в пиридине. Растворы инкубировали при 37°C в течение 90 минут с встряхиванием с интервалами 15 минут, затем добавляли 15 мкл N, O-бис- триметилсилилтрифторацетамида+триметилхлорсилана (BSTFA+TMCS) (99:1), затем раствор снова инкубировали при 37°C в течение 30 минут с встряхиванием с интервалами 15 минут, а затем добавляли и перемешивали 5 мкл смеси алканов (н-додекана, н-пентадекана, н-октадекана, н-эйкозана, н-пентакозана, н-гептакозана, н-дотриаконтана по 0,029% мас./об. каждого). Каждую дериватизирующую смесь выдерживали при температуре окружающей среды в течении 60 мин перед анализом GC-MS.
Анализ GC-MS метаболитов проводили на спектрометре Shimadzu TQ8050 газовой хроматографии с тандемной масс-спектрометрией, оборудованном капиллярной колонкой DB-5 (30 м×0,25 мм внутренний диаметр×толщина пленки 1 мкм). Один мкл впрыскивали в колонку с делением потока 1:10 с нагретым до 280°C впускным отверстием, и гелием в качестве газа-носителя. Температуру печи устанавливали на 100 °C, выдерживали в течение 4 минут, затем повышали до 320°C со скоростью 10 °C/минуту и выдерживали в течение 11 минут. Интерфейс масс-спектрометра нагревали до 280 °C, а источник ионов до 200 °C. Массы от 45 до 600 измеряли в режиме полного сканирования. Для режима селективной регистрации избранных реакций распада нескольких ионов (MRM) использовалась, для обнаружения с теми же параметрами GC и MS, библиотека Shimadzu MRM, содержащая 467 соединений с ионами-мишенями и ионами-спецификаторами между конкретными интервалами времени удерживания, установленными для каждого производного метаболита. Параметры селективной регистрации избранных реакций распада нескольких ионов (MRM) были разработаны для гомоцитриновой кислоты 4TMS и включены в протокол анализа MRM путем сканирования m/z=287, 243, 147 и 73 при энергии столкновения 3-45 В. На основе сканирования использовали следующие два спектра фрагментации для обнаружения при индексе удерживания 1931: ионы-мишени m/z=287> 73 при 21 вольте и ионы сравнения m/z=287/243 при 9 вольтах. Чтобы предотвратить загрязнение шприца для инъекций от одного образца к другому, шприц промывали по пять раз гексаном, а затем раствором этилацетата и ацетона в соотношении 1: 1 об./об., а затем промывали пиридином для удаления остатков гомоцитриновой кислоты 4TMS из предыдущего образца. Предполагаемые соединения, идентифицированные в режиме MRM, сравнивали с хроматограммой, полученной в режиме полного сканирования, где масс-спектр при конкретном времени удерживания сравнивали с библиотекой NIST 17 library и Golm metabolome database (Hummel et al., 2007).
Результаты продуцирования гомоцитрата в растительных клетках
Этот способ позволил легко обнаружить и измерить гомоцитрат во многих образцах. Контрольные образцы листьев N. benthamiana, которые были инфильтрированы только конструкцией p19 без последовательности NifV/HCS, показали низкие фоновые уровни гомоцитрата. Способ GC-MS/MS чрезвычайно чувствителен и поэтому неудивительно, что был обнаружен низкий уровень гомоцитрата. Сигнал в контрольных растениях считался подлинным, поскольку в способе использовались два диагностических иона, а время удерживания соответствовало аутентичному серийному стандарту. Фоновый шум для этих конкретных ионов отсутствовал только в стандартных смесях для контроля качества (QC) или в буфере для экстракции.
Неинфильтрированные образцы листьев и листья, инокулированные геном, кодирующим GFP, также показали низкие фоновые уровни гомоцитрата. Выбирали площадь пика до базовой линии, которая имела наибольшую площадь пика из трех негативных регуляций (GFP, p19, дикий тип). Для каждого образца, инфильтрированного геном NifV/HCS, площадь пика до базовой линии иона-мишени гомоцитрата вычиталась из площади пика для исследуемого образца. Нормализованные площади пиков были преобразованы в шкалу log10, а данные представлены на Рисунке 23.
Данные показали, что полипептиды NifV/HCS из K. oxytoca (KoNifV) и все три из Methanocaldococcus infernus (MiHCS1, MiHCS2 и MiHCS3) не продуцировали обнаруживаемого гомоцитрата выше базового уровня как для митохондриально-нацеленных полипептидов, так и для полипептидов нацеленных на цитоплазму. Эти данные согласуются с наблюдаемой нерастворимостью KoNifV и MiHCS для митохондриально-нацеленных полипептидов (Рисунок 20 и 21), но оставалось загадкой отсутствие активности HCS для полипептидов нацеленных на цитоплазму. Полипептиды M. infernus могли быть неактивны при температуре роста N. benthamiana. Напротив, и митохондриально-нацеленные слитые полипептиды, и слитые полипептиды, нацеленные на цитоплазму, содержащие семь других последовательностей NifV/HCS, явно были активны в продуцировании гомоцитрата в клетках листа.
Особого внимания заслуживают несколько специфичных наблюдений. Полипептиды HCS (ScHCS) S. cerevisiae были наиболее активными из исследованных полипептидов в продуцировании гомоцитрата, будучи в 10-100 раз более активными, чем другие полипептиды, независимо отмитохондриальной или цитоплазматической локализации. N-концевое продолжение из 22 аминокислот (последовательность scar), имеющее последовательность ISTQVVRNRGGYPYDVPDYAGG (SEQ ID NO:166), включая последовательность эпитопа НА на N-конце ScHCS, явно допустимо для функции HCS. Более короткая последовательность scar из 12-ти аминокислот MYPYDVPDYAGG(SEQ ID NO:165) на N-конце ScHCS также является допустимой для функции. Наиболее удивительно то, что слитой полипептид AvNifV из 406 аминокислот из SN254 (кодирующей MTP::HA::AvNifV, процессированный до scar9-HA::AvNifV) продуцировал в 27 раз больше гомоцитрата, когда он был нацелен на митохондрии растения по сравнению с полипептидом нацеленным на цитоплазму. Это также верно, но в меньшей степени, для HCS (CtHCS) Chlorobaculumtepidum, полученного из SN253. Вероятная причина этого заключалась в том, что полипептиды AvNifV и CtHCS были в некоторой степени чувствительны к кислороду, а локализация в митохондриях более защищена от кислорода и тем самым обеспечивает большую активность. В то же время определенное продуцирование гомоцитрата, когда AvNifV и CtHCS были локализованы в цитоплазме, предполагало, что эти два полипептида могли в некоторой степени переносить кислород. Данные о кислородной чувствительности AvNifV приведены в работе Zheng et al. (1997). Аналогичным образом HCS Thermincola potens, Thermoanaerobacter brockii и Methanosarcina acetivorans также продуцировали гомоцитрат независимо от локализации. Примечательно, что эти три HCS были более активными, когда локализовались в цитоплазме, что указывает на их слабую чувствительность к кислороду. Как это наблюдалось с ScHCS, продолжение из 22 аминокислот, имеющее последовательность ISTQVVRNRGGYPYDVPDYAGG(SEQ ID NO:166) на N-конце AvNifV, также было допустимо для функции.
ScHCS имел самый высокий уровень продуцирования гомоцитрата и поэтому был сочтен изобретателями наиболее подходящим NifV/HCS для использования в качестве части пути рекомбинантного Nif в митохондриях растения, если требуется продуцирование высокого уровня гомоцитрата. Однако любая из других последовательностей HCS может быть использована для синтеза FeMo-ко, поскольку гомоцитрат образует часть кофактора, который не используется в реакции нитрогеназы, и для этого потребуется немногое. Оптимальный уровень функции NifV можно определить эмпирически, как дополнительно описано в примерах ниже.
Синтез FeMo-ко и последующая активность нитрогеназы могут быть достигнуты in vitro путем комбинирования NifB, NifX, NifE, NifN, NifH, апо-NifD-NifK, NafY, Mo, Fe, S, s-аденозилметионина, регенерирующей смеси ATP (ATP, фосфокреатин, креатинфосфокиназа) и R-гомоцитрат, см работу Curatti et al. (2007), в которой предполагается, что NifV не нуждается во физическом взаимодействии с другими компонентами Nif, если в комбинированной смеси содержится достаточное количество гомоцитрата. Считается, что NifH, в частности, функционирует как ATP-зависимая Mo-гомоцитрат-инсертаза для доставки Mo-гомоцитрата в комплекс NifE-NifN для сборки FeMo-ко (Hu et al., 2013). Авторы изобретения посчитали, что если и существует возможный пагубный эффект от продуцирования высоких уровней гомоцитрата с помощью ScHCS в митохондриях растения, то AvNifV больше подходит в качестве части пути рекомбинантного Nif, чем ScHCS, поскольку фермент AvNifV с большей вероятностью доставляет произведенный им гомоцитрат в NifH путем физической ассоциации.
Измерение α-кетоглутарата и пирувата в клетках N. benthamiana.
Анализ GC-MS метаболитов также обнаружил производные α-кетоглутаровой кислоты (αKG) 1MEOX 2TMS и пировиноградной кислоты 1MEOX 1TMS, производные αKG и пировиноградной кислоты, соответственно. αKG и ацетил-кофермент A (Ac-CoA), который продуцируется в результате окисления пирувата пируватдегидрогеназой, являются двумя субстратами, ферменты NifV/HCS которых используются для синтеза гомоцитрата. Когда ScHCS экспрессировался в листьях N. benthamiana, он снижал уровень αKG и пирувата по сравнению с тем, когда NifV A. vinelandii был нацелен на митохондрии, который был по существу таким же, как и в листьях негативной регуляции без NifV/HCS. Поскольку αKG и пируват являются ключевыми промежуточными продуктами цикла TCA в митохондриальном матриксе, то снижение их уровней может оказывать пагубное влияние на общую функцию митохондрий, поэтому следует избегать чрезмерной экспрессии NifV/HCS до вредных уровней. Таким образом, был сделан вывод, что AvNifV лучше подходит для сборки FeMo-ко, FeV-ко или FeFe-ко, чем ScHCS, если гомоцитрат, продуцированный AvNifV, может быть доставлен к NifH посредством белок-белкового взаимодействия, а не путем диффузии, которая, возможно, требует более высокой концентрации гомоцитрата.
Пример 16. Растворимость вариантов NifH при экспрессии в растительных клетках
Введение
Было обнаружено, что полипептид NifH из Klebsiella oxytoca (KoNifH; SEQ ID NO:1) в основном нерастворим, а в некоторых экспериментах полностью нерастворим в митохондриях растения при экспрессии в виде слитого полипептида с последовательностью MTP и последовательностью эпитопа HA в системе для транзиентной экспрессии листьев (Пример 3). Был сделан вывод, что слитой полипептид NifH некорректно укладывается в митохондриях N. benthamiana или остается связанным с мембранами, даже несмотря на то, что последовательность MTP была правильно расщеплена MPP, и поэтому маловероятно, что он будет функционировать должным образом как белок NifH в этой ситуации. Напротив, соответствующий слитой полипептид NifH, включающий последовательность NifH K. Oxytoca , но без последовательности MTP на N-конце, нацеленной в цитоплазму, а не в митохондрии, был растворим в системе для транзиентной экспрессии листьев. Таким образом, нерастворимость слитого полипептида связана с митохондриальной локализацией. Ранее в работе Lopez-Torrejon et al. (2016) отмечали, что NifH из Azotobacter vinelandii сохраняет функцию переноса электронов NifH и растворим в митохондриях дрожжей. Однако на конференции в Стокгольме, Швеция, было заявлено, что NifH из A. vinelandii накапливался только с низким уровнем при экспрессии в митохондриях растения в системе для транзиентной экспрессии листьев, предположительно из-за слабой растворимости (Xi Jiang at ENFC in Stockholm, 2018). Изобретатели отметили, что дрожжевые клетки и растительные клетки могут различаться в отношении растворимости и/или функции любого специфичного полипептида NifH.
Результаты исследования
В попытке обойти проблему очевидной нерастворимости слитых полипептидов KoNifH, которые были исследованы на высокий уровень экспрессии в N. benthamiana, изобретатели, используя описанный ниже процесс, провели поиск гомологов белков NifH из других организмов, которые могли бы быть растворимы в качестве слитых полипептидов в митохондриях растения.
Последовательности полипептида NifH были извлечены из базы данных InterPro с использованием семейства IPR005977 - железосодержащий белок NifH нитрогеназы - в качестве запроса, а доступ к базе данных был получен 23 апреля 2018 года. Аминокислотные последовательности NifH в количестве 4183 штук были идентифицированы и извлечены из базы данных. Для выбора и исследования репрезентативных последовательностей была создана белковая сеть на основе сходства белков, что привело к кластеризации полипептидов NifH на основе сходства последовательностей. Для этого аминокислотные последовательности были выровнены с помощью ПО MAFFT (Программа множественного выравнивания аминокислотных или нуклеотидных последовательностей ), версия 7 с использованием сервера mafft.cbrc.jp/alignment/server/large.html?aug31. Использовали стратегию G-large-INS-1 для менее чем 10 000 последовательностей, короче чем 5 000 сайтов. Выходные данные были преобразованы из формата.pir в формат.phy с помощью онлайн-конвертера последовательностей (www.hiv.lanl.gov/content/sequence/ FORMAT_CONVERSION/form.html). Для вычисления матриц расстояний и подготовки данных во входных файлах для Cytoscape, была использована программа PHYLIP/protdist для вычисления матрицы расстояний Кимуры для последовательностей NifH. Выходной файл изменяли в блокноте Notepad++, чтобы подготовить соответствующий входной формат для aMATReader в ПО Cytoscape. Затем матрицу расстояний изменяли в Excel для уменьшения размера файла и определения подгрупп: все значения, превышающие 0,1, были удалены, в результате чего были созданы подгруппы, избыточные последовательности были удалены, нулевые значения были удалены, и значения были округлены до трех знаков после запятой.
Эта матрица расстояний была импортирована в Cytoscape с помощью приложения aMATReader в качестве неориентированной сети, с использованием ограничителя табуляции, для отмены выбора строк для импорта. На этом этапе сеть содержала 3114 узлов и 450 489 ребер графа. Сеть визуализировали с использованием алгоритма Prefuse Force Directed Layout (невзвешенный). Дополнительная информация была извлечена из базы знаний UniProt, включая название статьи, статус, названия белков, названия генов, организм, длину и таксономическое происхождение (PHYLUM), и импортирована в Cytoscape. Узлы были раскрашены по типу, а узлы, представляющие последовательности, выбранные для биохимического анализа, отображались как более крупные узлы. Белковые последовательности длиной более 700 аминокислот были удалены из сети, было удалено одиннадцать белковых последовательностей, так как длина этих последовательностей (731-804 аминокислотных остатка) не соответствовала длине типичного белка NifH на уровне 260-300. аминокислотных остатков. Девять из 11 последовательностей представляли собой вид Methanosarcina, одна вид Anaerovirgula multivorans и одна вид Treponema azotonutricium. Эти белки обычно обозначались как «NifEH». Первая часть каждого NifEH имела последовательность, аналогичную NifH, включая P-петлю, сайт связывания кластера [Fe4S4], а вторая часть каждой последовательности была связана с NifE или NifD, соответственно. У вида Methanosarcina есть ген, кодирующий NifD или аналогичный полипептид, локализованный рядом с геном NifEH, но отсутствует эквивалент NifK, локализованный в непосредственной близости. Эти полипептиды NifEH могут иметь различную функцию, даже если они структурно родственны белкам нитрогеназы. Насколько известно изобретателям, такие белки не упоминались в научной литературе, а экспериментальные данные отсутствуют.
Окончательная сеть содержала 3103 узла и 450 486 ребер графа. Для создания и визуализации сети использовали Cytoscape версий 3.6.1 и 3.7.0. База данных InterPro не содержала отдельных семейств для белков AnfH или VnfH, поэтому они были включены в группу NifH. Содействующие сигнатуры из баз данных участников InterPro, а именно CDD, TIGRFAMs и HAMAP, не делали различий между NifH, AnfH и VnfH. Поэтому последовательности AnfH и VnfH также были включены в процесс выравнивания. Подмножество последовательностей AnfH было идентифицировано из последовательностей NifH.
Выбор последовательности
Представителя каждой кластерной группы, содержащей более 13 последовательностей, выбирали для биохимического анализа для сравнения с NifH K. oxytoca. Это охватывало последовательности NifH изтермофильныхазотфиксирующих организмов, которые должны были быть исследованы на растворимость и функциональный анализ (Таблица 17). В столбце для температуры в Таблице 17 указывается оптимальная температура роста для некоторых организмов. Степень идентичности последовательности SEQ ID NO:1, каждой из выбранных последовательностей NifH приведена в Таблице 18. Аминокислотные последовательности слитых полипептидов, содержащих выбранные полипептиды NifH, отличные от KoNifH и слитые на С-конце последовательности MTP-CoxIV-TwinStrep, представлены в SEQ ID NO:168-181.
Таблица 17: Последовательности NifH из азотфиксирующих и родственных организмов, исследованные на растворимость в ходе экспрессии в качестве слитых полипептидов в листьях N. benthamiana
Khumanthem et al. (2007).
50-70 (опт. 65)
Штамм Carboxydothermus pertinax, вероятно, не был способен к азотфиксации, поскольку кодирующая белок NifD область в этом организме имела внутренний терминирующий кодон. Следовательно, у последовательности NifH также могла отсутствовать функциональность.
Таблица 18: Идентичность аминокислотной последовательности по отношению к NifH из K. oxytoca (SEQ ID NO:1)
Исследование белков NifH на растворимость в системе для транзиентной экспрессии листьев
Растворимость различных полипептидов NifH при экспрессии в виде слитых форм MTP-CoxIV::TwinStrep::NifH для определения митохондриальной локализации растений оценивали в ходе анализа вестерн-блот с использованием антитела Strep. Последовательность TwinStrep помещали между последовательностями MTP и NifH. Этот эпитоп, при необходимости, использовали для последующей очистки слитых полипептидов NifH. Белковые экстракты получали из инфильтрированных тканей листа и фракционировали в аэробных условиях на растворимые и нерастворимые фракции. Растворимость каждого слитого полипептида NifH оценивали после совместной экспрессии с NifM K. oxytoca, экспрессированного из генетической конструкции SN44 (кодирующей MTP-FAγ51::NifM::HA), чтобы убедиться в возможности увеличения растворимости в результате совместной экспрессии с NifM. Считается, что NifM участвует в созревании NifH в K. oxytoca. Неизвестно, требуется ли полипептидам NifH, из других исследованных видов, NifM-подобный белок для полной активности. Большинство этих организмов, кроме протеобактерий, не содержат гомолога NifM в своем геноме, однако другие негомологичные белки могут выполнять аналогичную функцию вместо NifM.
Анализ вестерн-блот (Рисунок 24) показал, что растворимый или, по меньшей мере, частично растворимый белок NifH был обнаружен в слитых полипептидах, включающих последовательности NifH из M.luminosus, M. infernus, H. modesticaldum, C. tepdium, Geobacter sp. M21 и M. thermoautotrophicus. В растворимой фракции, содержащей NifH из K. oxytoca, A. brasilense, F. casurinae, M. gracile и B. diazoefficans, было обнаружено немного слитого полипептида NifH или его отсутствие. Был сделан вывод, что большинство полипептидов NifH, которые были, по меньшей мере, частично растворимы в митохондриях растительной клетки, были получены из термофильных бактерий, возможно, потому, что такие полипептиды по своей природе были более стабильны, чем полипептиды из мезофильных бактерий, и поэтому они могли бы легче укладываться и сохраняться в их естественных конформациях.
Также было замечено, что отсутствовало значительное увеличение растворимости, когда слитой полипептид NifH был совместно экспрессирован с NifM K. oxytoca, в том числе с NifH K. oxytoca (Рисунок 24). Как упоминалось выше, было неизвестно, требовали ли белки NifH у большинства исследованных видов бактерий активности, подобной NifM, для созревания и продуцирования полностью функционального NifH.
Очистка NifH из M. infernus и NifH из M. laminosus из листьев N. benthamiana
Слитые полипептиды Twin Strep::NifH для NifH из M. infernus и M. lauminosus были успешно очищены из инфильтрированных образцов листьев N. benthamiana после экстракции при неденатурирующих условиях и затем с использованием колонки StrepTactinXT. Это подтвердило, что MPP-процессированные слитые полипептиды NifH этих двух видов действительно растворимы в митохондриях клеток листа. Очищенные белки используются для биохимического анализа, например, анализа на наличие кластеров FeS и на способность полипептидов NifH отдавать электроны очищенному NifD-NifK, выделенному из A. vinelandii.
Исследование вариантных полипептидов NifH в бактериальной системе нитрогеназы
Кандидаты NifH, которые оказались растворимыми при нацеливании на митохондрии листа, были исследованы на функцию NifH в E. coli с использованием системы MIT2.1. Сайт XhoI вводили в 3’-конец кодирующей белок NifH области в MIT2.1 посредством сайт-направленного мутагенеза. Этот недавно введенный сайт рестрикции XhoI вместе с существующим сайтом XhoI перед NifH использовали для отдельной замены последовательности NifH K. oxytoca дикого типа в MIT2.1 семью выбранными вариантными последовательностями NifH, которые были синтезированы с сайтами рестрикции XhoI, фланкирующими каждую открытую рамку считывания: M. laminosus NifH (MlNifH; Genbank Учетный №. Q47917), M. infernus NifH (MiNifH; Genbank Учетный №. WP_013099459), H. modesticaldum NifH (HmNifH; Genbank Учетный №. WP_012282218), C. tepidum NifH (CtNifH; Genbank Учетный №. WP_010933198), Geobacter sp. NifH (GspNifH; Genbank Учетный №. WP_015837436), M. thermautotrophicus NifH (MtNifH; Genbank Учетный №. AAB86034), и Carboxydothermus pertinax NifH (CpNifH; Genbank Учетный №. WP_075859892). Замену вариантов NifH проводили с pHJ-TOPO, содержащим NifHDKYENJ K. oxytoca, затем с pB-ori, содержащим вторую половину NifBQFUSVWZM из MIT2.1 K. Oxytoca,, и затем лигировали с модифицированным pHJ-TOPO после того, как обе плазмиды были расщеплены SbfI.
Полученные модифицированные плазмиды MIT2.1, содержащие вариантные NifH, использовали для трансформации штамма JM109 E. coli и трансформантов, исследуемых при анализе восстановления ацетилена. По сравнению с JM109 с исходным MIT2.1 в качестве позитивной регуляции в ARA, ни один из штаммов JM109, несущих модифицированные плазмиды MIT2.1 с вариантными NifH, не снижал ацетилен, вместо этого показывая тот же фоновый уровень продуцирования этилена, что и JM109 в плазмиде pB-ori в качестве негативной регуляции. Исходя из этого, изобретатели пришли к выводу, что вариантные NifH не будут функционировать с белками NifD-NifK из K. oxytoca, но будут функционировать с их соответствующим гетеротетрамером NifDK, например, NifH C. tepidum с NifD-NifK C. tepidum. Таким образом, совместимость каждого NifH с NifD-NifK можно определить эмпирически.
Пример 17. Экспрессия NifH и NifM в стабильно трансформированных растениях
Введение
Функциональный белок NifH, также известный как белок Fe, необходим для активности нитрогеназы. Он выполняет несколько известных функций для активности нитрогеназы: он необходим для передачи электронов ферменту нитрогеназы; для созревания металлокластеров, включая P-кластер; и участвует в синтезе кофакторов FeMo-ко, FeV-ко и FeFe-ко для Mo-нитрогеназы, V-нитрогеназы и Fe-нитрогеназы, соответственно. Ранее Rubio et al., осуществлили совместную экспрессию NifH и NifM Azotobacter vinelandii в дрожжах с митохондриальным нацеливанием. Очищенный белок NifH из дрожжевых клеток был способен отдавать электроны in vitro в голо-комплекс NifD-NifK (Lopez-Torrejon, 2016), но другие функции полипептида NifH не исследовались в той системе in vitro, которая не была предназначена для этого. Введение полностью функционального NifH в органеллы, например, митохондрии растения, будет иметь важное значение для инженерии нитрогеназы в растения.
Ранее изобретатели, используя транзиентную систему в листьях N. benthamiana, показали, что NifH (KoNifH) Klebsiella oxytoca может быть нацелен на митохондрии растений путем трансляционного слияния последовательности MTP с N-концом полипептида KoNifH (Allen et al, 2017). Слитой полипептид хорошо экспрессируется и процессируется расщеплением внутри последовательности MTP, демонстрируя локализацию экспрессированного слитого полипептида в митохондриях. При трансляционном слиянии на N-конце KoNifH два разных митохондриально-нацеливающих пептида MTP-FAγ77 и MTP-FAγ51, эффективно и специфично расщеплялись в предполагаемом сайте в MTP. Содержание процессированного слитого полипептида NifH было относительно высоким по сравнению с другими митохондриально-нацеленными белками Nif. Кроме того, эксперименты, описанные в данном документе в Примере 4, продемонстрировали, что «последовательность scar» из 9-ти аминокислот от С-конца последовательности MTP, оставшаяся после расщепления протеазой митохондриального матрикса (MPP) с дополнительным линкером Gly-Gly, а всего 11 аминокислот на N-конце KoNifH не снизили активность восстановления ацетилена при анализе в формате бактериальной комплементации.
Однако в случае слитого полипептида MTP-FAγ51::KoNifH::HA (SEQ ID NO:25), кодированного векторами SN18 и SN27,MPP-расщепленный полипептид scar9::KoNifH::HA обнаруживали исключительно в пределах нерастворимой белковой фракции (Примеры 2 и 3). Для оценки, может ли нерастворимость NifH быть следствием нацеленного пептида, была создана и исследована другая генетическая конструкция (SN42) с использованием другой последовательности MTP, кодирующей MTP-CoxIV::TwinStrep::KoNifH::HA (SEQ ID NO:128). Хотя коректно процессированную форму, полученную из MTP-CoxIV::TwinStrep::KoNifH::HA, легко обнаруживали при анализе транзиентной экспрессии листьев после расщепления в предполагаемом сайте в MTP, однако этот продукт scar32::KoNifH::HA обнаруживали преимущественно в нерастворимой белковой фракции.
Поскольку нерастворимые белки, вероятно, укладываются с нарушениями или остаются связанными с мембранами и, следовательно, нефункциональными, изобретатели искали различные альтернативные способы повышения растворимости NifH, описанные ниже. Функциональные последствия N-концевого продолжения из 32 аминокислот к KoNifH также были исследованы, что соответствует полипептиду, продуцированному после расщепления слитого полипептида MTP-CoxIV::TwinStrep::KoNifH::HA с помощью MPP в митохондриях растения.
Генетические и биохимические исследования с Azotobacter vinelandii и Klebsiella oxytoca показали, что NifM необходим для продуцирования функционального и зрелого белка NifH в этих азотфиксирующих бактериях. Как показано в Примерах 2-4 в данном документе, митохондриально-нацеленную версию NifM, MTP-FAγ51::KoNifM::HA (SEQ ID NO:123), экспрессировали, корректно и эффективно расщепляли в митохондриях растения и обнаруживали в растворимой фракции. Однако последовательность scar из 9 аминокислот на N-конце KoNifM снижала активность восстановления ацетилена только до 10-20% от уровня дикого типа в системе MIT2.1 E. coli (Таблица 4). Протеомный анализ бактериальных штаммов, продуцирующих процессированный полипептид scar9::NifM::HA, показал, что это N-концевое добавление к NifM привело примерно к 50-кратному увеличению накопления модифицированного полипептида NifM по сравнению с NifM дикого типа. Поскольку известно, что активность нитрогеназы чувствительна к изменениям уровней экспрессии различных белков Nif (Temme et al., 2012), то существует вероятность, что избыток scar9::NifM в этом формате бактериального анализа снижал функцию нитрогеназы до 10-20% по сравнению с уровнем дикого типа.
Совместная экспрессия NifH и NifM в листьях N. benthamiana
Для исследования возможности увеличения растворимости NifH в митохондриях растения в результате совместной экспрессии с слитым полипептидом NifM, который также был нацелен на эту органеллу, смесь штаммов Agrobacterial, каждый из которых содержал другой вектор, была инфильтрирована в листья N. Benthamiana, как описано в Примере 1. Смешивали и инфильтрировали первый штамм, который содержал либо вектор, кодирующий MTP-FAγ51::KoNifH::HA (SN18), либо вектор, кодирующий слитой полипептид MTP-CoxIV::TwinStrep::KoNifH::HA (SN42), и второй штамм, который содержал SN30, кодирующий слитой полипептид MTP-FAγ51::KoNifM::HA. Через пять дней после инфильтрирования из тканей листа получали фракции общего, растворимого и нерастворимого белка и подвергали их анализу вестерн-блот. Постоянного увеличения растворимости полипептида NifH в этих комбинациях, по сравнению с инфильтрированием одним вектором, не наблюдалось.
В качестве дополнительной попытки был сконструирован другой вектор, который имел два гена на одной и той же T-DNA, один ген, кодирующий слитой полипептид MTP-CoxIV::TwinStrep::KoNifH::HA (SEQ ID NO:128), и другой ген, кодирующий полипептид MTP-FAγ51::HA::KoNifM (SEQ ID NO:167). Первый ген имел эпитоп TwinStrep между последовательностями MTP и NifH и эпитоп HA на С-конце. Второй ген имел эпитоп НА между последовательностями MTP и NifM. Генетическую конструкцию с двумя генами обозначали SL6. Ее создавали с использованием модульной системы сборки DNA, известной как метод сборки Golden Gate, как описано выше. Ген, кодирующий слитой полипептид KoNifH, находился под контролем усиленного промотора 35S, тогда как ген, кодирующий полипептид KoNifM, представлял собой промотор SCSV S4 (Учетный №. AY181084).
Культуры Agrobacterium, трансформированные SL6, инфильтрировали в листья N. benthamiana. Образцы собирали через пять дней после инфильтрирования и получали фракции общего, растворимого и нерастворимого белка. Анализ вестерн-блот белковых экстрактов показал, что совместная экспрессия слитых полипептидов NifH и NifM из одного и того же вектора стабильно не увеличивала растворимость NifH, хотя, по меньшей мере, один эксперимент показал увеличение содержания растворимого полипептида NifH.
Затем было решено использовать SL6 для трансформации N. tabacum (табак) и N. benthamiana для продуцирования стабильно трансформированных растений с интегрированной в ядерный геном растения T-DNA.
Протокол трансформации растения
Для трансформирования растения N. benthamiana, растения выращивали в культуре клеток ткани в асептических условиях в качестве источника растительного материала для трансформации. Исходные растения получали из поверхностно-стерилизованных семян. Для этого семена промывали 70% этанолом, затем стерилизовали поверхность 5% гипохлоритом натрия в течение 10 минут при перемешивании и с последующим промыванием несколькими сменами воды. Затем семена проращивали на чашках, содержащих среду MSO при 4,43 г/л (M519, PhytoTechnology Laboratories), содержащую 3% сахарозы и 0,8% агара, при pH 5,8. Растения выращивали в вегетационном домике при 26°C с использованием светового периода 16/8 часов. Примерно через 2 недели развивающиеся всходы переносили в глубокую чашку с культурой клеток ткани и прореживали до 4 всходов, а затем культивировали в той же среде и в тех же условиях роста. Примерно через 2 недели отдельные укоренившиеся растения культивировали в горшках с культурой клеток ткани. Листья 6-ти недельных растений N. benthamiana использовали для трансформации, опосредованной Agrobacterium.
Культуры штамма AGL1 A. tumefaciens, содержащие генетические конструкции в бинарных векторах, например, SL6, выращивали при 28°C в среде MG/L с антибиотиками для поддержания отбора генетических конструкций. Культуры, имеющие оптическую плотность, составляющую от 0,25 до 0,5 при длине волны 600 нм, использовали для инокуляции тканей N. benthamiana следующим образом. Верхние листья растений, выращенных к культуре клеток ткани, вырезали и помещали в среду MG/L для сохранения упругости до использования и разрезали на части площадью примерно 1 см2, включая средние жилки листьев. Культуру Agrobacterium, содержащую генетическую конструкцию, добавляли к частям листа, чтобы эксплантаты были полностью влажными, и оставляли на 20-30 минут при периодическом встряхивании, чтобы позволить бактериям связаться с растительными клетками по краям среза. Затем инокулированные эксплантаты слегка промокали стерильной фильтровальной бумагой для удаления избытка Agrobacteria и переносили адаксиальной стороной вниз в среду без антибиотиков для совместного культивирования MS9. MS9 содержала среду MSO с 3% сахарозы, 0,8% агара при pH 5,8, стерилизованную автоклавированием, и растительные гормоны IBA, в концентрации 1 мг/л, и IAA, в концентрации 0,5 мг/л, добавленные после автоклавирования и охлаждения агаровой среды MSO до 55 °C. Инокулированные эксплантаты совместно культивировали при 26°C в течение 48 ч в темноте. После периода совместного культивирования эксплантаты переносили в среду для регенерации побегов (MS9 и растительные гормоны IBA в концентрации 1 мг/л и IAA в концентрации 0,5 мг/л плюс канамицин в концентрации 100 мг/л и тиментин в концентрации 150 мг/л) адаксиальной стороной вверх, высевая около 10 эксплантатов на чашку. Эксплантаты инкубировали при 26°C с использованием светового периода 16/8 часов. Эксплантаты переносили в свежую среду для регенерации побегов каждые 2-3 недели, пока не произошло развитие побегов. Через 6-8 недель побеги, которые развились до достаточного размера, переносили в среду для инициации роста корней (⅓MSO+100 мг/л канамицина+150 мг/л тиментина+1 м/л IBA). После появления у отдельных растений хорошо развитых корней, собирали небольшие образцы листьев для экстракции DNA и исследования с помощью PCR на наличие селектируемого маркерного гена и требуемых трансгенов. Трансгенные растения затем высаживали в почву и выращивали в теплице, позволяя растениям постепенно акклиматизироваться.
Растения Nicotiana tabacum сорта Wisconsin 38 (Wi38) трансформировали стандартными способами (Horsch et al., 1985).
Двенадцать независимо трансформированных растений были получены с помощью SL6 в N. benthamiana, обозначенных SL6 1-12, и еще двенадцать растений были получены в N. tabacum, SL6 13-24. Эти исходные трансгенные растения были обозначены как поколение T0. Наличие T-DNA в каждом из растений подтверждали с помощью PCR для DNA, полученной из образцов листьев растений, верифицируя, что все растения были трансгенными. Эти независимо трансформированные растения выращивали до созревания, а семена Т1 собирали после самоопыления каждого растения. Для исследования сегрегации трансгена в одной линии, 60 семян Т1 растения, обозначенного SL6-13, высевали в почву и выращивали в течение четырех недель в стандартных условиях теплицы. Наличие трансгенов оценивали с помощью PCR. Двадцать растений не имели трансгена (нуль-сегреганты), а 40 растений показали на PCR положительный результат, что указывает на трансгенный объект с малым числом копий, вероятно, имеющий одну вставку T-DNA в растение SL6-13. Несколько нуль-сегрегантов были идентифицированы и сохранены в качестве негативной регуляции.
Продуцирование слитых полипептидов NifH и NifM в трансгенных растениях оценивали экстракцией общего белка и обнаружением, с использованием в анализе вестерн-блот либо антитела анти-Strep, либо антитела анти-HA. Уровень слитого полипептида NifH в стабильно трансформированных растениях табака был намного ниже, чем уровни, наблюдавшиеся ранее при транзиентной экспрессии в листьях N. benthamiana. С удивлением и неожиданно, принимая во внимание более ранние результаты этих экспериментов, растения табака, включая растение, обозначенное SL6-13, продуцировали обнаруживаемые уровни корректно процессированного NifH, который был обнаружен исключительно в растворимой фракции. Аналогичным образом, растения N. benthamiana, стабильно трансформированные SL6, продуцировали значительно меньше полипептида NifH, однако этот полипептид подвергался эффективному процессингу и также обнаруживался в растворимой фракции.
Анализ потомства трансгенных растений
Листья разного возраста собирали с растений-потомков, чтобы определить какое-либо влияние возраста листьев на накопление, процессинг и растворимость слитых полипептидов NifH и NifM. Образцы отбирали из двух растений N. tabacum, которые были потомками SL6-13, причем с каждого растения был взят молодой лист, лист «среднего возраста» и более старый лист. В каждом из листьев, слитой полипептид NifH подвергали анализу вестерн-блот с использованием антитела анти-Strep, а в каждом из листьев полипептид NifM анализировали с антителом анти-НА. Уровень накопления слитого полипептида NifH увеличивается с возрастом листьев.
Очистка слитого полипептида NifH из стабильно трансформированных растений
Учитывая, что полипептид NifH с меткой TwinStrep, был растворим и что было достаточное количество растительного материала, то этот полипептид очищали с использованием аффинной среды StreptactinXT. Приблизительно 90 г материала из листьев растений SL6-13 экстрагировали путем гомогенизации материала в неденатурирующем буфере, подвергали центрифугированию для удаления продуктов распада клеток и фильтрации через фильтры 0,22 мкм, и пропускали через колонку StreptactinXT. После элюирования из колонки с использованием биотина, фракции, содержащие полипептид NifH, собирали и концентрировали. Образцы анализировали с помощью протеомики и подвергали анализу вестерн-блот с антителом анти-Strep для обнаружения полипептида NifH и антитела анти-HA для обнаружения обоих полипептидов NifH и NifM (Рисунок 25). Очищенный белок подвергали N-концевому анализу для определения аминокислотной последовательности на N-конце. Эти анализы подтвердили, что CoxIV MTP был расщеплен в предполагаемом сайте расщепления MPP. Очистка NifH путем связывания в колонке StreptactinXT также подтвердила вывод о растворимости TwinStrep::KoNifH, экстрагированного из стабильно трансформированных растений. В целом, эти результаты показали, что белок scar32::TwinStrep::KoNifH::HA, выделенный из стабильно трансформированных растений N. tabacum, корректно процессировался в митохондриях и является полностью растворимым, что соответствует двум основным требованиям для функции NifH в растениях.
Совместная экспрессия NifS и NifU с NifH и NifM в трансформированных растениях N. benthamiana
Генетические конструкции, кодирующие слитые полипептиды NifS (SN31) и NifU (SN32), инфильтрировали в растения N. benthamiana, трансформированные SL6, чтобы определить, может ли совместная экспрессия слитых полипептидов NifS и NifU повышать уровень накопления полипептидов NifH.
Пример 18. Экспрессия полипептидов Anf в растительных клетках
Введение
Система только железосодержащей нитрогеназы, обнаружена у некоторых диазотрофных бактерий, например у A. vinelandii, который имеет три системы нитрогеназы: систему на основе молибдена (Mo), систему на основе ванадия (V) и систему на основе только железа (Fe) с использованием кофакторов FeMo-ко, FeV-ко и FeFe-ко, соответственно (Davis et al., 1996; Robson et al., 1986). Ферменты как молибденовой нитрогеназы (Mo-нитрогеназа), так и ванадиевой нитрогеназы (V-нитрогеназа), которые фактически катализируют восстановление молекулярного азота, имеют известные кристаллические структуры. Хотя кристаллическая структура только железосодержащей нитрогеназы (Fe-нитрогеназа) еще не установлена, однако считается, что она имеет структуру, аналогичную структуре ванадиевой нитрогеназы (Sippel & Einsle, 2017). Все зарегистрированные на сегодняшний день организмы, содержащие одну или обе V- или Fe-нитрогеназы, также содержат Mo-нитрогеназу. Как правило, V- и Fe-нитрогеназы подавляются экспрессией Mo-нитрогеназы и экспрессируются только тогда, когда доступность Mo становится ограниченной. Для различения нитрогеназы молибденового типа от альтернативной нитрогеназы, можно использовать анализ изотопного восстановления ацетилена (ISARA), который измеряет изотопы 13C в ходе анализа восстановления ацетилена (Zhang et al., 2016).
Fe-нитрогеназа наименее изучена из этих трех систем. Она имеет самую низкую каталитическую активность нитрогеназы среди трех систем, но ее биогенез, по-видимому, проще, требуя меньше белков для активности нитрогеназы. Существует 6 известных белков Fe-нитрогеназы из хорошо изученного организма Azotobacter vinelandii, а именно AnfD, AnfK, AnfH, AnfG, AnfO и AnfR, которые отличаются от Fe-нитрогеназы. Известно, что из этих 6 белков первые 4 белка необходимы для активности и вносят вклад в активность фермента нитрогеназы. Каждая система нитрогеназы требует каталитических белков, обозначенных как Nif (или Vnf или Anf) D, K и H, а Fe-нитрогеназа использует белки AnfD, AnfK и AnfH. V- и Fe-нитрогеназам также требуется дополнительный структурный белок, обозначенный VnfG или AnfG, соответственно, который не требуется Mo-нитрогеназе. Гены anfO и anfR локализованы ниже других структурных генов anf, но их функция неизвестна, и было показано, что они не влияют на активность Fe-нитрогеназы при экспрессии в системе E. coli. (Yang et al., 2014). Оставшееся минимальное количество дополнительных генов, необходимых для активности Fe-нитрогеназы, является общим для Mo-нитрогеназного пути, а именно NifS, NifU, NifB, NifV, NifJ и NifF (Yang et al., 2014). Следовательно, только железосодержащая нитрогеназа имеет минимальный набор из 4 Anf и 6 дополнительных полипептидов Nif, необходимых для гетерологичной функции внутри E. coli (Yang et al., 2014).
В системе Fe-нитрогеназы фермент динитрогеназа, который является сайтом восстановления молекулярного азота, представляет собой гетерогексамер, состоящий из двух полипептидов AnfD в качестве α-единицы, двух полипептидов AnfK в качестве β-единицы и двух полипептидов AnfG в качестве δ-единицы, поэтому в конформации представляет собой α2β2δ2. Фермент редуктазы динитрогеназы, облигатный донор электронов фермента динитрогеназы, представляет собой гомодимер с 2 идентичными полипептидами AnfH. Редуктаза динитрогеназы также известна как белок Fe и содержит единственный кластер [Fe4S4] на границе раздела его субъединиц (Burén, Young, et al., 2017). Также предполагается, что белок AnfH будет выполнять две другие функции, в том числе он необходим для созревания фермента динитрогеназы аналогично продуктам генов NifH и VnfH в Mo- и V-нитрогеназах.
Что касается Mo- и V-нитрогеназ, то инжиниринг растений для экспрессии Fe-нитрогеназы считается чрезвычайно трудным. Все ключевые ферменты нитрогеназы требуют особой биохимической среды, чрезвычайно чувствительны к кислороду и требуют большого количества ATP, источника восстановителя, а такие элементы, как Fe, Mo, V и S, должны быть доступны в достаточных количествах в правильных участках клетки. В частности, ферменты Anf быстро необратимо инактивируются при воздействии кислорода. Как упоминалось выше, в растению необходимо ввести минимальный набор из 4 полипептидов Anf и 6 дополнительных полипептидов Nif, что очень сложно сделать с технической точки зрения.
Поэтому были проведены эксперименты по экспрессии генов Anf в растительных клетках с целью митохондриальной локализации продуктов гена Anf, как описано ниже. Поскольку 4 ключевых белка Anf представляют собой белки AnfD, AnfK, AnfH и AnfG, то изобретатели сначала исследовали 4 генетические конструкции, каждая из которых экспрессирует отдельный ген Anf, а затем объединили 4 гена в одну T-DNA в одном векторе.
Конструкции единичного гена для экспрессии слитых полипептидов Anf в растительных клетках
Была разработана и изготовлена первая серия генетических конструкций для раздельной экспрессии полипептидов AnfD, AnfK, AnfH и AnfG в растительных клетках, таких как клетки листьев N. benthamiana. Каждый синтетический ген находился под контролем сильного промотора 35S и области полиаденилирования/терминатора транскрипции CaMV 3’, которые фланкировали кодирующую белок область. Последовательности Anf из A. vinelandii использовали для конструирования кодированных аминокислотных последовательностей, а нуклеотидные последовательности были кодон-оптимизированы для экспрессии в растительных клетках. Для митохондриальной локализации, конструкции кодировали слитые полипептиды, содержащие MTP-FAγ51, слитой с N-концом, и либо эпитоп HA, либо эпитоп TwinStrep для обнаружения полипептидов с помощью анализа вестерн-блот с антителами анти-HA или анти-Strep, соответственно. Эпитоп НА был трансляционно слит либо с С-конца, либо, в большинстве случаев, между последовательностями MTP и Anf, тогда как эпитоп TwinStrep был слит с С-концом последовательностей Anf. Для каждой генетической конструкции, кодировавшей митохондриально-нацеленный слитой полипептид, были также созданы две соответствующие контрольные конструкции. Первая конструкция кодировала полипептид, у которого отсутствовала последовательность MTP и, следовательно, меньше экспрессировала полипептид нацеленный на цитоплазму, который обеспечивал сравнение молекулярной массы в ходе анализа вестерн-блот для MPP-процессированного полипептида из полипептидов MTP-Anf (процессированного Anf), с подтверждением того, что MPP-процессированный полипептид в каждом случае включает «последовательность scar» примерно из 9 аминокислот, и поэтому идентичность размеров не сохранялась. Вторая контрольная конструкция в каждом случае кодировала слитой полипептид, который имел 13 аминокислот в последовательности MTP, замещенных аланинами (Allen et al., 2017), предназначенных для предотвращения MPP-процессинга. Таким образом, эти вторые контрольные полипептиды обеспечивали сравнение молекулярной массы для непроцессированного полипептида из соответствующей конструкции MTP-Anf. Последовательность MTP с мутацией аланина была обозначена здесь как mFAγ51. После анализа белковых экстрактов из инфильтрированных тканей растения, образец из каждой конструкции MTP-Anf и двух его соответствующих контрольных конструкций загружали на соседние дорожки для гель-электрофореза, что позволило наилучшим образом обнаруживать процессинг полипептида MTP-Anf. Впоследствии предполагаемый сайт расщепления в мотиве MTP подтверждали масс-спектрометрией.
В случае необходимости сохранения функции AnfK для слитых полипептидов, имеющих последовательность AnfK, удаляли С-концевые продолжения, по сравнению с полипептидом дикого типа. Необходимость использования С-концевой последовательности дикого типа для AnfK была аналогична использованию С-конца дикого типа для NifK из K. oxytoca (WO2018/141030), поскольку С-концевые продолжения отменяли функцию (Yang et al., 2017).
Конструкции с одним геном перечислены в Таблице 19, в которой также перечислены предполагаемые молекулярные массы (кДа) каждого полипептида до и после процессинга в митохондриях с помощью MPP. В Таблице 19 также перечислены SEQ ID NO для непроцессированных слитых полипептидов. Генетические конструкции создавали методами сборки Golden Gate аналогично конструкциям, описанным в предыдущих Примерах.
Согласно Таблице 19, контрольными конструкциями для конструкций AnfD (SN81 и SN161) были конструкции: SN82, которая продуцировала полипептид, приблизительно соответствующий размеру процессированной формы; и конструкция SN158, которая продуцировала полипептид, имеющий размер непроцессированной формы. Таким образом, экстракты белков из этих конструкций обрабатывали на соседних дорожкам на стадии гель-электрофореза в ходе анализа вестерн-блот. Для конструкции AnfK SN129 контрольными конструкциями были SN152 и SN155. Для конструкции AnfH SN130 контрольными конструкциями были SN153 и SN156. Для конструкции AnfG SN131 контрольными конструкциями были SN154 и SN157.
Помимо изменения позиции эпитопа НА на С-конце или по направлению к N-концу была сделана еще одна вариация в одной конструкции (SN195), заключающаяся в использовании последовательности MTP CoxIV (Burén et al., 2017) вместо последовательности MTP-FAγ51.
Таблица 19. Конструкции единичного гена для экспрессии слитых полипептидов Anf в растительных клетках. (NA: Не применяется)
Экспрессия слитых полипептидов Anf в клетках листа N. benthamiana
Каждую из конструкций отдельно вводили в растения N. benthamiana с помощью способов, опосредованных Agrobacterium, как описано в Примере 1. Образцы листьев собирали через 4-5 дней после инфильтрирования, получали и анализировали белковые экстракты электрофорезом в полиакриламидном геле в присутствии додецилсульфата натрия (PAGE) и анализом вестерн-блот, как описано в предыдущих примерах. Таким образом, экспрессированные полипептиды исследовали на импорт в митохондриальный матрикс в результате MPP-процессинга лидерных последовательностей MTP. В дальнейших экспериментах белковые экстракты фракционировали на растворимые и нерастворимые фракции, используя способ, описанный в Примере 1.
Когда неочищенные белковые экстракты подвергали анализу вестерн-блот с использованием антитела анти-НА, легко обнаруживались полипептидные полосы, которые соответствовали предсказанным размерам полипептидов Anf (Рисунок 26). Все отдельные митохондриально-нацеленные полипептиды, содержащие последовательности AnfD, AnfK, AnfH и AnfG, хорошо экспрессировались и были видны после короткой экспозиции (2 мин) в ходе анализа вестерн-блот. Каждая из конструкций SN161, SN130 и SN131 для слитых полипептидов AnfD, AnfH и AnfG, соответственно, имеющая последовательность MTP-FAγ51, давала одну доминантную полосу на мембране для блоттинга с молекулярной массой, ожидаемой для полипептида, процессированного MPP в пределах последовательности MTP. Полосы на соседних дорожках для контрольных полипептидов в каждом случае подтверждали, что эти полосы были для процессированных полипептидов. Был сделан вывод, что эти три слитых полипептида хорошо экспрессируются и эффективно процессируются в митохондриях. Впоследствии процессинг подтверждали масс-спектрометрией. Образец из SN130, кодирующий MTP-FAγ51::HA::AnfH, также показал менее четкую, но, тем не менее, определенную полосу с более высокой молекулярной массой, размером, подходящим для димера полипептида, несмотря на условия денатурации белка, используемые во время стадии гель-электрофореза.
Дорожки для двух конструкций AnfK были более сложными с несколькими полосами. Митохондриально-нацеленные и нацеленные на цитоплазму полипептиды AnfK, продуцированные из SN152 и SN129, соответственно, показали дополнительные полосы, обнаруживаемые антителом HA, которые были меньше, чем ожидалось для расщепления в последовательности MTP, что указывает на то, что полипептид AnfK, по-видимому, подвергается дополнительному протеолитическому расщеплению. Полипептиды меньшего размера, примерно на 4-6 кДа меньше по размеру, также могут возникать в результате преждевременной терминации транскрипции или трансляции. Несмотря на это наблюдение для AnfK, был сделан вывод, что все четыре генетические конструкции, включая N-концевую последовательность MTP, экспрессировали предполагаемый слитой полипептид с процессингом, частично в случае AnfK, для образованиялокализованных в митохондриях требуемых полипептидов Anf.
Экспрессия и процессинг слитых полипептидов Anf в растительных клетках из мультигенных конструкций
В первом, описанном выше эксперименте, использовали конструкции с одним геном для получения отдельных слитых полипептидов Anf. Теперь изобретатели решили исследовать экспрессию всех четырех слитых полипептидов AnfD, AnfK, AnfH и AnfG из одного вектора, где каждый из генов Anf имеет свой собственный промотор 35S и терминатор транскрипции. Этот эксперимент был направлен на исследование каких-либо взаимодействий между четырьмя полипептидами Anf после их совместной экспрессии в одних и тех же растительных клетках, в частности, на поиск изменений в уровнях накопления отдельных полипептидов или в их MPP-процессинге. Для этого собирали генетическую конструкцию из бинарного вектора, содержащую все четыре гена в одной T-DNA, причем каждый ген имел последовательность MTP-FAγ51, трансляционно слитую с эпитопом НА, за которой следовала последовательность Anf. Использовали те же нуклеотидные последовательности и аминокислотные последовательности, что и для векторов SN161, SN129, SN130 и SN131 с одним геном. Полученную генетическую конструкцию обозначали SL26. Были также изготовлены две контрольные конструкции: конструкцию SL31, которая кодировала четыре слитых полипептида Anf, каждую последовательностью MTP с мутацией аланина для продуцирования маркеров размера для непроцессированных полипептидов (mFAγ51::HA::Anf); и конструкцию SL36, которая кодировала четыре слитых полипептида без последовательности MTP (HA::Anf) в качестве маркера размера для процессированных полипептидов. Кроме того, для упрощения идентификации множественных полипептидных полос в ходе анализа вестерн-блот, были созданы три дополнительных вектора путем поэтапной делеции одного, двух или трех генов из SL26: В SL27 был удален ген AnfG, в SL28 были удалены гены AnfH и AnfG, а в SL29 были удалены гены AnfK, AnfH и AnfG, и оставлен только ген AnfD. Мультигенные векторы и составляющие их гены перечислены в Таблице 20.
Все эти мультигенные векторы по отдельности вводили в листья N. benthamiana способом, описанным в Примере 1. Белки экстрагировали из тканей листа через 4 или 5 дней после инфильтрирования и подвергали анализу вестерн-блот как и ранее. Результаты (Рисунок 27) показали, что все четыре полипептида Anf, слитые с последовательностями MTP-FAγ51 и HA, легко обнаруживаются и хорошо экспрессируются в виде отдельных интенсивных полос. Кроме того, слитые полипептиды AnfD, AnfH и AnfG, имеющие N-концевые лидерные последовательности MTP-FAγ51, эффективно процессировали в последовательности MTP, а слитой полипептид AnfK подвергали частичному процессингу, о чем свидетельствует сравнение с размерами соответствующих полипептидов, экспрессированных из SL31 на соседней дорожке. Это было подтверждено в отдельном эксперименте с использованием мультигенной конструкции SL36, которая кодировала 4 полипептида HA::Anf, у которых отсутствовала последовательность MTP, и таким образом продуцировала маркеры размера в ходе анализа вестерн-блот для процессированных полипептидов из SL26. Анализ вестерн-блот экстрактов, полученных из ряда векторов SL26, SL27, SL28 и SL29 (Рисунок 27, панель C), помог идентифицировать четыре полипептида в смеси, подтвердив их идентичность, как и в смеси четырех векторов с одним геном в полосе с отметкой Mix.
Уровни накопления четырех полипептидов Anf можно сравнивать при экспрессии из мультигенной конструкции по отношению к смеси из конструкций с одним геном. Для мультигенной конструкции слитой полипептид AnfD накапливался на более высоком уровне, чем три других полипептида Anf (Рисунк 27, панель A), что было неожиданно, учитывая, что ген NifD был наиболее трудно экспрессируемым для Mo-нитрогеназы среди соответствующих генов NifD, NifK и NifH (Allen et al., 2017). Кроме того, полипептид AnfD оказался полноразмерным, и отсутствовало доказательство наличия скрытого сайта вторичного расщепления в AnfD, в отличие от наблюдений с NifD из K. oxytoca (Примеры 6 и 7).
Таблица 20. Перечень мультгенных векторов и кодированных полипептидов для экспрессии слитых полипептидов Anf в клетках растения
Подтверждение митохондриальной локализации и процессинга MPP
Для авторов изобретения, процессинг слитых полипептидов MTP-FAγ51::HA::Anf был четким указанием на митохондриальную локализацию четырех процессированных полипептидов Anf, экспрессированных из SL26. Это дополнительно подтверждалось обогащением митохондриальных фракций, полученных из инфильтрированных тканей листа, с использованием опосредованного метаксином способа, как описано в Примере 13. Это включало добавление генетической конструкции SN197, кодирующей слитой полипептид с метаксином последовательности распознавания TwinStrep-mTurquoise-TEV (SEQ ID NO:121) в A. Tumafaciens в смеси с Agrobacterium, содержащей SL26. Область метаксина полипептида из SN197 при транзиентной экспрессии в растительных клетках локализовалась на внешней мембране митохондрий(Lister et al., 2007). Это экспонировало N-концевой мотив TwinStrep в цитозоль, обеспечивая быструю очистку меченых митохондрий в мягких условиях с использованием магнитных микроносителей, покрытых антителом анти-Strep. Это привело к значительному обогащению митохондриальных белков по сравнению с немитохондриальными белками в той же клетке.
Для исследования, смесь культур A. tumefaciens, содержащая SN197 в одном штамме и SL26 в другом штамме, была введена в листья N. benthamiana. Инфильтрированные тканей собирали через 5 дней. Эти ткани процессировали для выделения митохондрий, как описано в Примере 13. Затем белки в выделенных митохондриях подвергали электрофорезу SDS-PAGE и анализу вестерн-блот использованием антитела HA для обнаружения. Все полипептиды Anf легко обнаруживались в митохондриальной фракции. Полосы, обнаруженные в ходе анализа вестерн-блот, соответствовали размерам процессированных полипептидов AnfD, AnfK, AnfH и AnfG из SL26, что вновь указывает на то, что полипептиды Anf локализованы в митохондриях растения. Меньшая полоса из слитого полипептида AnfK, которая, вероятно, была образована дополнительным протеолитическим расщеплением (см. выше), также была обогащена в митохондриальной фракции, что предполагает проявление вторичного расщепления в митохондриях. Наблюдение за процессингом полипептидов Anf свидетельствует о том, что они локализованы в митохондриальном матриксе.
Процессинг расщеплением в последовательности MTP был подтвержден способами LC-MS после триптического расщепления белков с использованием способов, описанных в Примере 1. Полосы белка выделяли из гелей, окрашенных кумасси, после электрофореза белковых экстрактов, экспрессированных из SL26. Идентичность белка в срезах геля подтверждали с помощью LC-MS и нацеленного MRM. Идентичность белков соответствовала метаксину, AnfD, AnfK и AnfH с достоверностью не менее 95%. Белок AnfG не был идентифицирован в геле кумасси, который подвергался обнаружению способом LC-MS, вероятно, из-за его низкого уровня накопления. Все белки Anf, за исключением AnfK, были обнаружены с предполагаемым N-концом после эффективного расщепления MTP. У полипептида AnfK, два N-концевых пептида-мишени FAγ51 MTP были обнаружены MRM при низком уровне сигнала, что указывает на частичное расщепление MPP слитого полипептида AnfK. Это согласуется с наблюдениями, сделанными с помощью анализа вестерн-блот, и подтверждает, что частичное расщепление происходит в предполагаемом сайте в последовательности MTP под действием MPP.
Растворимость митохондриальных и слитых полипептидов после экспрессии в растительных клетках
Авторы изобретения посчитали, что для сохранения функциональности белков Fe-нитрогеназы их необходимо процессировать в растворимой форме, чтобы обеспечить необходимые межбелковые взаимодействия и стабильность фермента Fe-нитрогеназы, а также позволить ферментам взаимодействовать со своими субстратами и кофакторами. Нерастворимость белков могло быть признаком неправильной укладки белка или плотного связывания с митохондриальными мембранами, что вредно для активности нитрогеназы. Поэтому были проведены эксперименты, чтобы исследовать, находятся ли экспрессированные полипептиды Anf в растворимой форме при их продуцировании в митохондриях растения. Это осуществляли путем фракционирования белковых экстрактов на растворимую (надосадочную жидкость) и нерастворимую (осадок) форму с использованием способа, описанного в Примере 1.
Впервые это было сделано, как и раньше, с использованием конструкций с одним геном в листьях N. benthamiana. Белковые экстракты растворимых и нерастворимых белков приготовляли из листьев, инокулированных генетическими конструкциями, и подвергали анализу вестерн-блот (Рисунок 28). Анализ вестерн-блот показал, что при нацеливании слитого полипептида AnfD в митохондрии в листьях N. benthamiana, он был практически нерастворим, а у растворимой фракции была видна только очень слабая полоса (Рисунок 28). Процессированные и непроцессированные полипептиды AnfK, по существу, присутствовали только в растворимой фракции, тогда как процессированный полипептид AnfH был растворим только частично. Митохондриально-нацеленный полипептид AnfG присутствовал только в растворимой фракции, что указывает на то, что процессированный слитой полипептид AnfG находился в растворимой форме при экспрессии с митохондриальным матриксом N. benthamiana.
Растворимость митохондриально-нацеленных AnfD, AnfK, AnfH и AnfG при совместной экспрессии исследовали следующим образом. Растворимость слитых полипептидов AnfD, AnfK, AnfH и AnfG, экспрессированных из мультигенного вектора SL26, сравнивали с растворимостью полипептидов, экспрессированных из SL31. Смесь штаммов Agrobacterium, каждый из которых содержит конструкцию с одним геном, также использовали для инфильтрироваия растений. Анализы вестерн-блот представлены на Рисунке 27, панель В.
Удивительный и неожиданный результат наблюдали с мультигенным вектором SL26. На этот раз часть процессированного слитого полипептида AnfD четко наблюдали в растворимой фракции, что указывало на то, что совместная экспрессия других полипептидов Anf увеличивала растворимость, по меньшей мере, некоторых полипептидов AnfD. Это свидетельствует о том, что либо часть полипептида AnfD была возможно стабилизирована за счет ассоциации белка с белком полипептида AnfD с одним или несколькими другими полипептидами Anf, либо увеличенная укладка полипептида AnfD происходила в своей соответствующей конформации. Возможность ассоциации белок-белок исследовали в соответствии со следующим Примером.
Серии векторов SL26, SL27, SL28 и SL29 использовали в аналогичном эксперименте для сравнения растворимости полипептида AnfD при совместной экспрессии с одним, двумя или всеми тремя другими полипептидами Anf. Слитые полипептиды, экспрессированные из мультигенных векторов SL26, SL27 и SL28, и вектора SL29 с одним геном исследовали на уровни накопления полипептидов и на наличие растворимых и нерастворимых полипептидов AnfD. Результаты транзиентной экспрессии листьев N. benthamiana показали, что по мере уменьшения количества генов Anf уменьшалась и растворимость полипептида AnfD, особенно в отсутствие AnfK. Таким образом, был сделан вывод, что присутствие AnfK, в частности, увеличивает растворимость полипептида AnfD.
Дальнейшее подтверждение растворимости митохондриально-локализованных полипептидов Anf было получено с помощью эксперимента по аффинной очистке с использованием магнитных микроносителей, связанных с антителом анти-НА. В том случае, когда неочищенные экстракты взаимодействовали с магнитными микроносителями, а несвязанные белки промывались перед анализом связанных белков, каждый из слитых полипептидов AnfD (как процессированные, так и непроцессированные формы), AnfK (как процессированные, так и непроцессированные формы), AnfH (процессированные) и AnfG (процессированные) выделяли из магнитных микроносителей. Полосы полипептидов, обогащенные НА, которые наблюдали на гелях, окрашенных Кумасси, вырезали, а полипептиды в срезах геля анализировали с помощью масс-спектрометрии LC-MS. Полосы, присутствующие в гелях, имели правильные размеры как для процессированных, так и для непроцессированных полипептидов AnfD, AnfK и AnfG. Полипептиды, идентифицированные для AnfG вероятно содержали частично процессированный полипептид, имеющий дополнительную аминокислоту на N-конце сайта расщепления MTP. Это согласуется с наблюдением двух близко мигрирующих полос, присутствующих для AnfG в анализе вестерн-блот для экстрактов из SL26 (см. Рисунок 27, панель C). Полоса для AnfH была только процессированного размера, что указывает на эффективный процессинг в последовательности MTP. Идентичность полипептидных полос подтверждали с помощью LC-MS.
Несколько других мультигенных векторов были сконструированы и изготовлены для исследования влияния позиции гена Anf в мультигенном векторе или позиции эпитопа НА на экспрессию белка и растворимость, или на то и другое вместе. Эти векторы включали конструкции, обозначенные SL23, SL30, SL34 и SL37. Разное позиционирование генов в мультигенном векторе, по-видимому, не оказывало значительного влияния на экспрессию и растворимость белка.
Вектор SL26 используется для трансформации растений табака (N. tabacum), N. benthamiana и Arabidopsis thaliana, продуцируя стабильно трансформированные растения, которые экспрессируют полипептиды AnfD, AnfK, AnfH и AnfG.
Обсуждение
Эти эксперименты продемонстрировали возможность экспрессии генов Anf, кодирующих слитые полипептиды AnfD, AnfK, AnfG и AnfH, и возможность их процессирования и локализации в митохондриях растительных клеток. Было доказано, что полипептиды расщепляются в предполагаемом сайте в последовательности MTP, оставляя в каждом случае «последовательность scar» из 9 аминокислот на N-конце слитого полипептида. Митохондриальная локализация также была продемонстрирована несколькими различными способами. Вводили одно- и многогенные конструкции и проводили анализ транзиентной экспрессии листьев N. benthamiana. Также исследовали растворимость митохондриально-локализованных полипептидов Anf. Растворимость AnfD увеличивалась при использовании мультигенных конструкций для совместной экспрессии AnfK, AnfH и AnfG.
Пример 19. Синергетическое взаимодействие полипептидов Fe-нитрогеназы в митохондриях листьев растений
Белки AnfD, AnfK и AnfG образуют гетерогексамерный комплекс, который, с необходимыми кофакторами, составляет фермент динитрогеназы (Davis et al., 1996; Zheng et al., 2018). Этот комплекс является каталитическим ферментом для восстановления молекулярного азота. Чтобы проявить себя активным ферментом, этот комплекс требует FeFe-кофактора и нескольких кластеров Fe-S.
Авторы изобретения разработали и провели несколько экспериментов для обнаружения белок-белковых взаимодействий полипептидов Anf в митохондриях растения после экспрессии из мультигенного вектора. Для исследования, в первом эксперименте, был разработан и изготовлен вектор, обозначенный SL30 (Таблица 20), который содержал гены anfD, anfK, anfH и anfG, каждый из которых экспрессировался своим собственным промотором 35S и с теми же терминаторами транскрипции, что и для SL26. Важной модификацией по отношению к SL26 было то, что слитой полипептид AnfD из SL30 имел эпитоп TwinStrep, слитой с С-концом AnfD, чтобы обеспечить очистку полипептида AnfD в мягких, неденатурирующих условиях. Для митохондриальной локализации, SL30 все еще имел последовательность MTP-FAγ51, слитую с N-концом AnfD. Слитые полипептиды AnfK, AnfH и AnfG, кодированные SL30, имели последовательность MTP-FAγ51, трансляционно слитую на N-конце полипептидов, за которой следует эпитоп HA, а затем последовательность Anf, как для SL26. Каждый отдельный ген в SL30 сохранял свой собственный промотор 35S и терминатор, как и в случае использования SL26.
SL30 вводили в A. tumefaciens и культуры трансформированных Agrobacteria, инфильтрированных в листья N. benthamiana, как и ранее. Через пять дней образцы листьев собирали и процессировали в условиях окружающей среды для экстракции растворимых белков в буфер для экстракции с использованием того же буфера для экстракции, что и в Примере 14. Смесь неочищенных белков пропускали через колонку для аффинной хроматографии Strep-Tactin XT в аэробных условиях. После промывки колонки 10 объемами промывочного буфера (как в Примере 14) для удаления несвязанных белков, связанные белки элюировали промывочным буфером, содержащим 50 ммоль биотина, pH 7,2, и подвергали электрофорезу SDS-PAGE и анализу вестерн-блот с использованием антитела Strep-Tactin для обнаружения полипептида AnfD, и антитела анти-НА для обнаружения любых совместно очищаемых полипептидов Anf, имеющих эпитоп НА.
Экстрагированные белки подвергали анализу вестерн-блот с использованием антитела Strep-tactin. Анализ показал, что очищенный полипептид AnfD присутствовал в элюате и что он мигрировал при молекулярной массе для процессированной формы (Рисунок 26), что указывало на то, что митохондриально-нацеленный AnfD подвергался процессингу, был растворим и взаимодействовал с аффинной средой Strep-tactin. При анализе вестерн-блот с антителом HA наблюдали слабую, но четко видимую полосу, соответствующую слитому полипептиду AnfK, мигрирующую со скоростью, соответствующей скорости правильно MPP-процессированной изоформы AnfK. Это указывает на то, что слитой полипептид AnfK был очищен совместно с полипептидом AnfD. Полипептид AnfG не был виден в ходе анализа вестерн-блот. На мембранах вестерн-блот также наблюдали несколько полос с более низкой молекулярной массой, которые могли представлять собой продукты распада AnfD, которые могли иметь место после экстракции.
Второй аналогичный эксперимент проводили таким же образом, за исключением того, что изготовили и использовали новую мультигенную конструкцию SL34. С помощью этой конструкции эпитоп TwinStrep сливали с последовательностью AnfK между последовательностями MTP и AnfK, а полипептид AnfD был аналогичен полипептиду, кодированному SL26 (Таблица 20), то есть с эпитопом НА. Эта конфигурация была разработана для исследования обратного захвата и обнаружения, по сравнению с экспериментом с SL26, в котором полипептид AnfK мог быть очищен в колонке Strep-Tactin, а связанные белки могли быть проанализированы с помощью антитела HA на наличие других полипептидов Anf. Полипептид AnfK, кодированный SL34, содержал лидерную последовательность CoxIV MTP, слитую на N-конце AnfK с Twin-strep, а не с MTP-FAγ51. Слитые полипептиды AnfD, AnfH и AnfG, кодированные SL34, каждый имел последовательность MTP-FAγ51, трансляционно слитую на N-конце полипептида, за которым следует эпитоп НА. Ранее было показано, что CoxIV MTP правильно нацеливает белки на митохондриальный матрикс в N. benthamiana (Burén et al., 2017).
Культуру A. tumefaciens, содержащую SL34, инфильтрировали в листья N. benthamiana, а образцы листьев собирали через 5 дней. Образцы ткани процессировали, используя те же экспериментальные условия для SL30, а полученные экстракты неочищенного белка пропускали через колонку Strep-Tactin для очистки полипептида AnfK, содержащего последовательность TwinStrep. Элюат из колонки снова подвергали электрофорезу SDS-PAGE и анализу вестерн-блот использованием как антитела HA, так и антитела Strep-Tactin для обнаружения полипептидов, имеющих эпитопы HA и TwinStrep, соответственно. Анализ вестерн-блот с антителом Strep-Tactin показал, как и предполагалось, наличие очищенного полипептида AnfK в элюате, а молекулярная масса полипептида соответствовала массе MPP-процессированной изоформы. Дополнительный анализ вестерн-блот с антителом HA показал наличие полипептида AnfD, что указывало на то, что полипептид AnfD был очищен совместно с полипептидом AnfK. Молекулярная масса AnfD соответствовала массе MPP-процессированной изоформы. АВ ходе анализе вестерн-блот вновь не наблюдали, AnfG, но он был обнаружен позже с низкой интенсивностью сигнала с помощью масс-спектрометрии LC-MS. Этот эксперимент, как и предыдущий эксперимент с SL30, продемонстрировал, что MPP-процессированные полипептиды AnfD и AnfK, нацеленные на митохондриальный матрикс растительных клеток, ассоциировались друг с другом.
Был собран другой мультигенный вектор SL37 (Таблица 20), который кодировал слитой полипептид, имеющий последовательности MTP-CoxIV и Twin-strep, слитые на N-конце AnfK, и слитые полипептиды AnfH и AnfG, имеющие последовательность MTP-FAγ51 трансляционно слитую на N-конце других полипептидов Anf, за которой следует эпитоп HA. Полипептид AnfD также имел MTP-FAγ51 MTP, трансляционно слитую с N-концом, тогда как эпитоп HA был трансляционно слит на С-конце последовательности AnfD. Эта конструкция была разработана для исследования ассоциирования полипептида AnfK с полноразмерным процессированным полипептидом AnfD или, возможно, с укороченным продуктом AnfD. На этот раз экстракцию и процессинг белка проводили в анаэробных условиях. Белковый экстракт пропускали через колонку для аффинной хроматографии Strep-Tactin XT в аэробных условиях. Затем элюат подвергали электрофорезу SDS-PAGE и анализу вестерн-блот с использованием антител HA и Strep-Tactin для обнаружения.
Анализ вестерн-блот с использованием антитела Strep-Tactin показал наличие процессированного полипептида AnfK. Кроме того, анализ вестерн-блот с использованием антитела HA показал полипептидные полосы, соответствующие по размеру как процессированным, так и непроцессированным полипептидам AnfD, и полосы с более низкой молекулярной массой, представляющими собой более мелкие продукты AnfD, которые, вероятно, образовались после экстракции. Полосы полипептидов с размерами для полипептидов AnfH и AnfG наблюдали в элюате, но они имели гораздо меньшую интенсивность, чем у AnfK или AnfD (Рисунок 26).
Элюаты, продуцированные из образцов SL34 и SL37, анализировали с помощью масс-спектрометрии LC-MS и нацеленного MRM. Пептиды из полипептидов AnfK, AnfD и AnfG были обнаружены в обоих элюатах, а пептиды из AnfH обнаруживали только после анаэробной экстракции.
В качестве негативной регуляции для вышеупомянутых экспериментов и для исследования специфичности обнаружения, в листья N. benthamiana был введен SL26, который кодировал полипептиды AnfD, AnfK, AnfG и AnfH, которые были слиты с MTP-FAγ51 и имели эпитоп НА на N-конце (Таблица 20). Ткани листа процессировали таким же образом в аэробных условиях как для SL30 и SL34 и как описано выше. Единственная полипептидная полоса, наблюдаемая в ходе анализа вестерн-блот белковых экстрактов из SL26 с использованием антитела Strep-Tactin, представляла собой относительно слабую фоновую полосу. Полосы полипептида AnfВ в элюате, в ходе анализа вестерн-блот с использованием антитела Strep-Tactin или антитела HA, отсутствовали. Этот контрольный эксперимент продемонстрировал, что полипептиды, содержащие эпитоп НА, наблюдаемый при анализе вестерн-блот, представляли собой специфичный результат ассоциации полипептидов AnfD и AnfK.
Обсуждение
Мультигенные конструкции и дифференциальное мечение эпитопов в этих экспериментах использовали для демонстрации ассоциации между слитыми полипептидами AnfD и AnfK, нацеленными на митохондриальный матрикс растения. Эти результаты показали, что существует возможность продуцирования нескольких полипептидов Anf и их локализацию в митохондриях растения. Эти эксперименты впервые продемонстрировали продуцирование отдельных белков Fe-нитрогеназы в эукариотической среде, особенно в митохондриях растения. Совместная экспрессия нескольких генов Anf из одного вектора приводила к увеличению растворимости полипептида AnfD, хотя он все еще оставался только частично растворимым.
При процессинге в аэробных условиях часть очищенного полипептида AnfD очищали совместно с полипептидом AnfK. Обратный эксперимент проводили в аэробных условиях, когда AnfK был трансляционно слит с эпитопом TwinStrep, а все другие полипептиды Anf были слиты с эпитопом HA. Некоторое количество белка AnfD очищали совместно с полипептидом AnfK, а также с небольшим количеством белка AnfG. Когда аналогичный эксперимент проводили в анаэробных условиях, то снова обнаруживали только небольшие количества белка AnfG, что указывало на то, что полипептиды AnfD, AnfK и AnfG взаимодействуют в растворимой фракции митохондрий с образованием комплекса. Обнаружение AnfG и AnfD вместе с AnfK во время его очистки указывало на трехстороннюю ассоциацию. Также было продемонстрирована совместная очистка AnfG с AnfD-AnfK в анаэробных условиях. Предсказанная структура нитрогеназы FeFe содержит полипептид AnfG, физически взаимодействующий с поверхностью AnfD (Sippel и Einsle, 2017; Zheng et al., 2018). Интересно, что когда экстракцию проводили в анаэробных условиях, то небольшие количества белка AnfH также обнаруживали в элюате.
В экспериментах с осаждением белок AnfG наблюдали в меньшем количестве, по сравнению с AnfD и AnfK. Полоса правильного размера для AnfG была видна после более длительной экспозиции. Меньшее содержание AnfG может указывать на отсутствие оптимального соотношения субъединиц для гетерогексамера Fe-нитрогеназы.
На основании этих экспериментов изобретатели пришли к выводу, что ассоциация полипептидов AnfD и AnfK и трехсторонняя ассоциация AnfD, AnfK и AnfG продемонстрировали возможность использования этих компонентов Fe-нитрогеназы в митохондриях растения для инженерии нитрогеназы.
Пример 20. Продуцирование трансляционного слияния между AnfD и AnfK, нацеленного на митохондрии растения
Хотя сообщения о кристаллической структуре Fe-нитрогеназы отсутствовали, было предсказано, что субъединицы AnfD, AnfK и AnfG Fe-динитрогеназы у азотфиксирующих бактерий, которые имеют Fe-нитрогеназу, находятся в стехиометрическом соотношении 1: 1: 1 (Hu & Ribbe, 2015; Zheng et al., 2018). Это соотношение для полипептидов AnfD, AnfK и AnfG может быть важным для оптимальной функции Fe-нитрогеназы и может влиять на растворимость компонента AnfD. Как описано в этом Примере, была разработана предсказанная структурная модель для Fe-нитрогеназы. Модель использовали для создания олигопептидного линкера подходящей длины для присоединения С-конца AnfD к N-концу AnfK и, таким образом, образования трансляционного слияния AnfD и AnfK. Длину линкера рассчитывали таким образом, чтобы обеспечить правильную укладку белкового комплекса на основе предсказанной структурной модели. Были созданы и исследованы генетические конструкции для экспрессии слитого полипептида. Слитой полипептид имел последовательность МТР для его локализации в митохондриальном матриксе.
Построение структурной модели Fe-нитрогеназы
Для создания слитого полипептида AnfD::linker::AnfK разработали гомологическую модель для комплекса AnfDKHG исходя из кристаллической структуры V-нитрогеназы A. vinelandii PDB ID: 5N6Y (Sippel and Einsle, 2017). Модель использовали, поскольку о кристаллической структуре Fe-нитрогеназы не сообщалось, а V-нитрогеназа считалась ближайшей в гомологии последовательностей. Гомологические модели создавали с использованием сервера SWISS-MODEL (swissmodel.expasy.org/) для каждого из полипептидов AnfD и AnfK A. vinelandii дикого типа (SEQ ID NO:216 и 217) с использованием соответствующих мономеров из PDB ID: 5N6Y α2β2-гетеродимера в качестве матриц. В модели AnfD отсутствовал С-концевой остаток из 31 аминокислоты у последовательности дикого типа (NSETLRQYTGGYDSVSKLREREYPAFERKVG, SEQ ID NO:197), а в модели AnfK отсутствовали две N-концевые аминокислоты (PH). Полноразмерный гетеродимер конструировали с использованием функции «Matchmaker» в ПО Chimera для наложения гомологических моделей AnfD и AnfK на нативный 5N6Y α2β2-гетеродимер, после чего вышеупомянутые недостающие остатки вручную добавляли в модель с помощью Discovery Studio 2018 (Dassault Systèmes BIOVIA, San Diego). Остаток из 31 аминокислоты на С-конце мономера AnfD добавляли в виде α-спиралей, чтобы использовать консервативный подход к общей длине этого участка. AnfD был на 36 остатков длиннее на С-конце, чем структура VnfD, на которой он был построен, поэтому невозможно с уверенностью сказать, какую конформацию примет эта дополнительная последовательность. Таким образом, при моделировании использовали самый короткий вариант, доступный для 31 аминокислоты, которые изначально не были сконструированы во время создания гомологической модели.
Полноразмерная модель гетеродимера α2β2 без кофакторов была подготовлена для молекулярной динамики с использованием модуля Xleap ПО AMBER18 путем сольватации в водяном боксе в режимах периодических границ (TIP3P, усеченный октаэдр, минимальное граничное расстояние 12,0 Å от растворенного вещества) и нейтрализации ионами Na+ (frcmod. ionjc tip3p). Систему подвергали минимизации энергии с помощью ПО Amber18 с использованием 25000 шагов наискорейшего спуска, за которыми следовали 25000 шагов сопряженного градиента, а затем 20 нс молекулярной динамики с использованием ПО AMBER18. Белок обрабатывали силовым полем ff14SB, а моделирование проводили при 298 К (множественный NVT) с использованием порогового значения 12,0 Å с дальнодействующими взаимодействиями, обработанными способом суммирования по Эвальду с использованием сетки частиц. Задачей моделирования было выявление потенциальных областей с высокой деформацией и любых других потенциально вредных особенностей, поэтому для этой задачи было достаточно 20 нс. Траекторию анализировали с помощью VMD (hwww.ks.uiuc.edu/). Указанные α-спирали, сконструированные для 31 остатка, добавленного к C-концу AnfD, сохраняли свою структуру на протяжении всей траектории, хотя существует предположение, что, возможно, это была их естественная конформация, хотя для дальнейшего подтверждения потребуются более обширные динамические симуляции. Добавленные остатки и линкеры релаксировали на ранней стадии моделирования без видимых неблагоприятных взаимодействий с остальной структурой.
На основе модели было предсказано, что пептидный линкер, соединяющий C-конец AnfD с N-концом AnfK, может создавать слитой белок, который сохраняет свою общую структуру и, следовательно, сохраняет свою функцию. Для моделирования использовали исходную линкерную пептидную последовательность из 16 аминокислот, обозначенную «линкер16», имеющую аминокислотную последовательность GGGSGGGSGGGSGGGS (SEQ ID NO:198), которая, как ожидается, даст неупорядоченный линкер. Гомологические модели предсказали, что олигопептид длиной, по меньшей мере, в 16 аминокислот может простираться на необходимое расстояние. Поэтому линкер из 16 аминокислот добавляли в расширенной конформации, а затем релаксировали с помощью ряда грубых оптимизаций геометрии в ПО Discovery Studio.
Координаты слитого димера AnfDK получали из последней рамки моделирования молекулярной динамики в течение 20 нс, и эту структуру накладывали с помощью PDB ID: 5N6Y для создания исходных позиций для гомологической модели AnfG, которая была создана с помощью ПО SWISS-MODEL с использованием в качестве матрицы мономера VnfG из 5N6Y. После того, как модель Anf (DKG)2 была построена, она была наложена на модель NifDKH из PDB ID: 1N2C для создания исходных позиций для гомологической модели AnfH, которые были сконструированы в ПО SWISS-MODEL с использованием мономера NifH из PDB ID: 1N2C в качестве матрицы. Перед молекулярной динамикой, проводимой, как описано выше, димерные структуры AnfG и AnfH вручную позиционировали немного в стороне от их границ раздела со структурами слияния AnfD-AnfK, чтобы уменьшить пространственные конфликты, возникающие из-за артефактов суперпозиции.
Аминокислотная последовательность синтетического слитого полипептида с линкером16 представлена как SEQ ID NO:199. Смоделированная структура представлена на Рисунке 29.
Для целей обнаружения эпитоп НА, имеющий последовательность YPYDVPDYA (SEQ ID NO:115), добавляли в середину линкера из 16 аминокислот, чтобы создать последовательность из 26 аминокислот ggggsgggsypydvpdyagggsgggs (SEQ ID NO:200), обозначенную в настоящем документе как «линкер26(HA)». Эпитоп НА не был включен в минимизацию или молекулярную динамику. В каждом случае слитой полипептид с этим «линкером26(HA)» между последовательностями AnfD и AnfK и соединяющий их, и без N-концевой последовательности MTP (SEQ ID NO:201) или MTP-FAγ51 (SEQ ID NO:202), MTP-CoxIV (SEQ ID NO:203), mFAγ51 (SEQ ID NO:204) или последовательности 6xHis (SEQ ID NO:205), слитой с N-концом слитого полипептида, предполагал наличие AnfD.
Полипептиды AnfK, AnfG и AnfH должны связываться правильно, без прогнозируемых побочных эффектов для нативной структуры. В этих конструкциях белок AnfG не включали в эту конструкцию линкера, поскольку как N-, так и C-концы AnfG вводили в толщу тканей близко к поверхности AnfD и, по-видимому, маловероятно, чтобы они выдерживали какие-либо продолжения линкера. Также было продемонстрировано, что и AnfG, и AnfK не переносят продолжения С-концевой аминокислоты(Yang et al., 2018), что согласуется с гомологической моделью Fe-нитрогеназы, разработанной, как описано выше.
Конструкции, использованные в данном Примере, обобщены в Таблице 21.
Таблица 21. Перечень генетических конструкций, использованных в этом Примере
Синтез и исследование генетических конструкции для экспрессии полипептида AnfD-линкер-AnfK в клетках растения
Последовательность DNA, кодирующая кодирующую белок область AnfD::Linker26(HA)::AnfK, была химически синтезирована и использована для создания набора генетических конструкций методами сборки Golden Gate с использованием аминокислотных последовательностей A. vinelandii для AnfD и AnfK. Для экспрессии растения кодирующая белок область была кодон-оптимизирована. Экспрессия гена, кодирующего слитой полипептид, в растительных клетках находилась под контролем промотора 35S и области Nos3’ последовательности полиаденилирования/терминатора транскрипции (Таблица 21). Для митохондриального нацеливания, последовательность, кодирующую MTP-FAγ51::HA, добавляли выше области кодирующей белок AnfD::линкер26(HA)::AnfK таким образом, что при транскрибировании и трансляции аминокислотные последовательности MTP и HA трансляционно сливались с полипептидом AnfD::линкер26(HA)::AnfK как единый продукт трансляции. Генетическую конструкцию, кодирующую этот слитой полипептид обозначали SN272. Аминокислотная последовательность полноразмерного слитого полипептида, кодированная SN272 представлена как SEQ ID NO:202. Был изготовлен второй вектор, обозначенный SN273, который кодировал идентичный полипептид, за исключением того, что последовательность MTP из гена CoxIV с последовательностью TwinStrep (Burén et al., 2017) была заменена на последовательность MTP-FAγ51. Аминокислотная последовательность полноразмерного слитого полипептида, кодированная SN273 представлена как SEQ ID NO:203. Чтобы использовать маркеры молекулярной массы для обнаружения процессинга продукта трансляции в митохондриях, были созданы две контрольные генетические конструкции. У первого (SN274) отсутствовала последовательность MTP-FAγ51, и поэтому его нацеливали на цитоплазму. У второго (SN275) имелась мутированная последовательность MTP-FAγ51, которая предотвращала расщепление с помощью MPP, обозначенная как mFAγ51. Аминокислотные последовательности слитых полипептидов, кодированных SN274 и SN275, представлены как SEQ ID NO:204 и 205.
Эти векторы вводили отдельно в листья N. benthamiana с использованием опосредованных Agrobacterium способов, описанных в Примере 1. В качестве дополнительного контроля, векторы, экспрессирующие отдельные белки Anf в различных комбинациях SL26, SL28, SN161 и SN129, также инфильтрировали в листья N. benthamiana. Ткани листьев собирали через 4 дня после инфильтрирования и процессировали для получения фракций общего, растворимого и нерастворимого белка, как описано в Примере 1. Полученные фракции белка подвергали электрофорезу SDS-PAGE и анализу вестерн-блот использованием антитела HA для обнаружения.
Анализ вестерн-блот показал, что все слитые полипептиды AnfD::linker26(HA)::AnfK легко обнаруживались во фракциях общего белка, выделенных из листьев N. benthamiana (Рисунок 30). Молекулярная масса основной полипептидной полосы каждой конструкции соответствовала предсказанному размеру полипептидов в диапазоне от 110 до 120 кДа (см. Таблицу 21). Прогнозируемый размер полноразмерного (непроцессированного) слитого полипептида MTP-FAγ51::HA::AnfD::линкер26(HA)::AnfK составлял примерно 118 кДа. Было предсказано, что процессированный полипептид после расщепления имел массу примерно 113 кДа, что позволяло отличить его от непроцессированного полипептида по их разной подвижности в гелях SDS-PAGE и в ходе анализа вестерн-блот. Молекулярная масса полипептида, обнаруженная в ходе анализа вестерн-блот (Рисунок 30), соответствовала массе контрольного полипептида, кодированного SN275, который представлял собой процессированную форму, что указывало на то, что полипептид MTP-FAγ51::HA::AnfD::линкер26(HA)::AnfK из SN272 эффективно импортировался в митохондрии и расщеплялся внутри клеток N. benthamiana. Аналогичным образом, полипептидная полоса, созданная из конструкции SN273, кодирующей слитой полипептид MTP-CoxIV::TwinStrep::AnfD::линкер26(HA)::AnfK, также, по-видимому, эффективно и правильно процессировалась. Указанныйслитой полипептид MTP-FAγ51::HA::AnfD::линкер26(HA)::AnfK из SN272 имел два эпитопа HA, тогда как слитой полипептид MTP-CoxIV::TwinStrep::AnfD::линкер26(HA)::AnfK из SN273 имел только один эпитоп, поэтому первый полипептид в ходе анализа вестерн-блот обнаруживали более эффективно.
Анализ вестерн-блот растворимых и нерастворимых фракций показал, что экспрессия митохондриально-нацеленного AnfD из SN161 сама по себе приводила к преимущественно нерастворимому полипептиду (Рисунок 31, панель A), с видимыми полосами только с очень слабой интенсивностью. Однако растворимость полипептида AnfD увеличивалась, когда тот же ген AnfD совместно экспрессировали с AnfK из SL28, и дополнительно улучшалась, когда ген AnfD совместно экспрессировали с AnfK, AnfH и AnfG из SL26. В каждом случае, когда гены AnfD и AnfK совместно экспрессировали, полипептиды AnfD и AnfK обнаруживали в разных количествах в растворимой фракции, несмотря на то, что гены экспрессировали из одной и той же T-DNA. Напротив, трансляционное слияние AnfD и AnfK в форме митохондриально-нацеленного слитого полипептида MTP::HA::AnfD::линкер26(HA)::AnfK, как в SN272 и SN273, обязательно обеспечивало идеальное стехиометрическое соотношение для полипептидов AnfD и AnfK, составлявшее 1: 1. Авторы изобретения пришли к выводу, что слитой полипептид, использующий линкерную последовательность, имел, по меньшей мере, данное преимущество по сравнению с экспрессией полипептидов из отдельных генов, даже когда два гена связывали в одной T-DNA.
Полипептиды, полученные в результате процессинга полипептидов MTP::HA::AnfD::линкер26(HA)::AnfK, экспрессированных из SN272 и SN273, обнаруживали как в растворимой, так и в нерастворимой фракциях экстрактов растений (Рисунок 31, панели A) и B)). Поскольку добавление митохондриально-нацеленных генов, экспрессирующих AnfH и AnfG, увеличивало растворимость митохондриально-нацеленного AnfD, то в дальнейших экспериментах митохондриально-нацеленный MTP::HA::AnfD::линкер26(HA)::AnfK будут совместно экспрессировать вместе с митохондриально-нацеленными AnfH и AnfG.
Полипептид, полученный в результате процессинга слитого полипептида MTP-FAγ51::HA::AnfD::линкер26(HA)::AnfK, очищали после экспрессии гена из SN272 с использованием эпитопа НА способом аффинной очистки. Очищенный белок подвергают анализу с помощью протеомики, чтобы подтвердить, что N-концевая последовательность соответствует ожидаемой, как при расщеплении MPP.
Генетическая конструкция SN272 представляет собой бинарный вектор, который подходит для получения стабильно трансформированных растений посредством трансформации, опосредованной Agrobacterium, с добавлением селектируемого маркерного гена. Ген, кодирующий слитой полипептид, вырезают и вставляют в бинарный вектор, содержащий подходящий селектируемый маркерный ген. После этого полученный вектор используют для получения стабильно трансформированных растений табака и N. benthamiana. Показано, что слитой полипептид экспрессируется и расщепляется в последовательности MTP в предполагаемом сайте с помощью MPP, и показано, что он присутствует в митохондриях. По меньшей мере, часть процессированного слитого полипептида присутствует в растворимой фракции.
Пример 21. Продуцирование белков Anf и Nif, необходимых для Fe-нитрогеназы в растительных клетках, с помощью митохондриального нацеливания
Введение
Сообщалось (Yang et al., 2014), что для образования Fe-нитрогеназы в бактерии E. coli требуется минимум десять генов, кодирующих белки Anf и Nif, а именно 4 структурных гена Anf, кодирующих полипептиды AnfD, AnfK, AnfH и AnfG, и 6 так называемых дополнительных генов Nif, кодирующих полипептиды NifV, NifS, NifU, NifJ, NifF и NifB. Последовательности полипептидов Anf были основаны на азотфиксирующей бактерии A. vinelandii, а для других полипептидов Nif на бактерии K. oxytoca. Экспрессия набора из десяти генов в E. coli продуцировала функциональную Fe-нитрогеназу, хотя и с низкой активностью (Yang et al., 2014).
Основываясь на данных, описанных в предыдущих Примерах в настоящем документе, показывающих продуцирование слитых полипептидов Anf и Nif в митохондриях растений в растворимой форме, авторы настоящего изобретения решили попытаться сконструировать растительные клетки для получения минимального набора генов для продуцирования Fe-нитрогеназы, нацеливающей продукты генов в митохондриальный матрикс в растительных клетках.
Результаты исследования
Набор генных продуктов, выбранный для серии экспериментов, включал полипептиды AnfD, AnfK, AnfG и AnfH на основе диазотрофа A. vinelandii (Av) и 6 белков Nif, а именно NifF, NifJ, NifS и NifU на основе K. oxytoca (Ко), NifV из A. vinelandii (AvNifV) и NifB из Methanocaldococcus infernus (MiNifB). Генетические конструкции были сконструированы и изготовлены для экспрессии полипептидов в листьях N. benthamiana с нацеливанием на митохондриальный матрикс посредством трансляционного слияния N-концевых последовательностей MTP, аналогично генетическим конструкциям, описанным в предыдущих Примерах. Нуклеотидные последовательности для экспрессии слитых полипептидов были кодон-оптимизированы для экспрессии в растительных клетках, как и ранее. Были использованы две разные последовательности MTP, а именно MTP-FAγ51 и MTP-CoxIV, для митохондриального нацеливания слитых полипептидов. Полипептиды, которые имели MTP-FAγ51, имели эпитоп HA, слитой либо на N-, либо на C-конце, тогда как полипептиды, которые имели последовательность MTP-CoxIV, имели эпитоп TwinStrep, вставленный между ней и полипептидом Anf/Nif. Для экспрессии в N. benthamiana каждый ген находился под контролем промотора 35S и области nos 3’ последовательности полиаденилирования/терминатора транскрипции. Эти нуклеотидные последовательности располагались выше и ниже каждой области, кодирующей белок, соответственно. Сборка конструкций по-прежнему проводилась методами сборки Golden Gate.
Мультигенные конструкции SL42 и SL43 создавали с использованием этих принципов и методов. Каждый вектор содержал по пять разных отдельных генов, связанных в одну T-DNA (Таблица 22). SL42 имел гены, кодирующие слитые полипептиды, которые включали последовательности KoNifS, KoNifU, KoNifJ, KoNifF и MiNifB, причем каждую со своими собственными последовательностями MTP и эпитопа, слитыми трансляционно. SL43 имел гены, кодирующие слитые полипептиды, которые включали последовательности AvAnfD, AvAnfK, AvAnfH, AvAnfG и AvNifV, причем каждую со своими собственными последовательностями MTP и эпитопа. Последовательность AvNifV выбирали из множества доступных последовательностей NifV на основе данных экспрессии, процессинга и растворимости, а также свидетельств о продуцировании гомоцитрата AvNifV, нацеленного на митохондрии растения, как описано в Примере 15.
Таблица 22. Одиночная и мультгенная генетическая конструкция, кодирующая компоненты Fe-нитрогеназы для экспрессии в растительных клетках
линкер26(HA)::AvAnfK
линкер26(HA)::AvAnfK
Продуцирование слитых полипептидов в растительных клетках
Культуры A. tumefaciens, содержащие SL42, инфильтрировали в листья N. Benthamiana возрастом 5-недель, как описано в Примере 1. Образцы листьев собирали через четыре-пять дней после инфильтрирования. Фракции общих, растворимых и нерастворимых белков экстрагировали следующим образом. Для исследования растворимости полипептидов, экспрессированных растением, ткань листа растирали в охлажденном на льду связывающем буфере для экстракции (100 ммоль Трис pH 8,0, 150 ммоль NaCl, 0,25 моль/л маннита, 5% (об./ об.) глицерина, 1% (об. об.) Tween® 20, 1% (мас./об.) PVP, только что добавленные 2 ммоль ТСЕР, 0,2 ммоль PMSF и 10 мкмоль/л леупептина) и переносили в микроцентрифужную пробирку. Образец центрифугировали при 20000 x g в течение 5 минут для разделения образца на растворимую (надосадочную) и нерастворимую (осадочную) фракции. Надосадочную жидкость переносили в чистую микроцентрифужную пробирку и снова центрифугировали при 20000 x g в течение 5 минут, для удаления любого оставшегося нерастворенного вещества. Нерастворимую фракцию промывали путем ресуспендирования осадка в 300 мкл буфера для экстракции с диспергированием повторными движениями пипетки и центрифугировали при 20000 x g в течение 5 мин, удаляя надосадочную жидкость. Эту стадию промывки повторяли еще дважды, удаляя любой оставшийся растворимый белок из нерастворимой фракции. Образцы затем подвергали электрофорезу SDS-PAGE и анализу вестерн-блот с использованием антител анти-HA и анти-Strep. Антитело анти-HA (моноклональное анти-HA, Sigma) разводили в соотношении 1:5000, а конъюгат антитела анти-Strep/HRP (конъюгат Strep-MAB, HRP, IBA) разводили в соотношении 1:10000.
Анализ вестерн-блот для SL42 (Рисунок 32) показал, что все пять полипептидов легко обнаруживались соответствующими антителами, каждый из которых демонстрировал полосы полипептидов, присутствующие во фракции растворимого белка. Слитой полипептид NifJ, по-видимому, полностью процессируется MPP, тогда как полипептиды NifU, NifS и NifF присутствуют как в процессированной, так и в непроцессированной формах, что указывает на менее эффективное расщепление с помощью MPP. Полипептиды NifJ, NifU, NifS, NifF и NifB присутствовали как в растворимой, так и в нерастворимой фракциях. Полипептид NifB, который был трансляционно слит на N-конце с последовательностью MTP-CoxIV-Twin-strep, был виден, когда для обнаружения использовали антитело анти-Strep (Рисунок 31, панель B).
Анализ вестерн-блот для SL43 (Рисунок 33) также показал, что все пять кодированных полипептидов легко обнаруживались соответствующими антителами, каждый из которых демонстрировал полосы у полипептидов, присутствующие во фракции растворимого белка. Важно отметить, что процессированные слитые полипептиды AnfD, AnfK и AnfH все наблюдались в растворимой фракции. Они также наблюдались в нерастворимой фракции, что указывает на частичную растворимость этих трех слитых полипептидов. Этот результат был значительно лучше, чем наблюдалось при экспрессии соответствующих генов из векторов с единственным геном. Слитые полипептиды AnfD, AnfG, AnfH и NifV, по-видимому, частично расщеплялись MPP, поскольку каждый из них показывает полосы для процессированных и непроцессированных форм. Слитой полипептид AnfK, по-видимому, эффективно процессировался.
Затем культуры A. tumefaciens, содержащие SL42 и SL43, смешивали и инфильтрировали в листья N. benthamiana, как описано ранее. Таким образом, в этом эксперименте были представлены все 4 гена AvAnf и все шесть генов Nif, т. е. комбинация из 10 генов. Неожиданный и значительный результат, наблюдавшийся в ходе анализа вестерн-блот (Рисунок 34), заключался в том, что все 10 полипептидов легко обнаруживались. Более того, все 10 полипептидов присутствовали в растворимой фракции, некоторые из которых демонстрировали эффективный процессинг с помощью MPP. Некоторые полипептиды были видны в виде 2 полос, причем верхняя полоса представляла непроцессированный полипептид, а нижняя полоса представляла MPP-расщепленный полипептид и демонстрировала импорт в митохондрии. Полоса непроцессированного полипептида была видна для белков AnfD, NifV, NifU и NifF, как и полоса предсказанного размера для расщепленных полипептидов.
Ассоциация AnfD и AnfK в растительных клетках
Мультигенные векторы SL43 и SL49 (Таблица 22) инфильтрировали в растения N. benthamiana возрастом 5-недель по отдельности и в комбинации. SL43 кодировал слитой полипептид, имеющий четыре отдельных гена, кодирующих полипептиды AnfD, AnfH, AnfG и NifV, последовательностью MTP-FAγ51 трансляционно слитой на N-конце полипептидов Nif, за которой следовал эпитоп НА, и пятый ген, кодирующий последовательности MTP-CoxIV и Twin-strep, слитые на N-конце AnfK. SL49 кодировал слитые полипептиды NifJ, NifF и NifU последовательностью MTP-FAγ51, трансляционно слитой на N-конце полипептидов Nif, за которой следовал эпитоп HA на C-конце, и слитой полипептид NifB, имеющий слитые MTP-FAγ51 и HA на N-конце. Конструкции разрабатывали для очистки продукта полипептида AnfK с использованием эпитопа Twin-Strep и для исследования возможности совместной очистки других белков Anf или Nif.
Экстракцию и процессинг белка из совместно инфильтрированных образцов растений проводили в анаэробных условиях. Белковый экстракт пропускали через колонку для аффинной хроматографии Strep-Tactin XT и затем элюировали. Образцы, собранные в процессе очистки полипептидов, подвергали электрофорезу SDS-PAGE и анализу вестерн-блот с использованием антител HA и Strep-Tactin для обнаружения.
Анализ вестерн-блот, проведенный с использованием антитела Strep-Tactin, показал присутствие процессированного слитого полипептида AnfK в каждой общей фракции, входящей фракции, осадочной фракции и фракции элюата, демонстрирующих полосы с более низкими молекулярными массами, потенциально представляющими собой более мелкие продукты, производные от AnfK, которые, вероятно, были продуцированы в результате постэкстракционной деградации из-за загрязнения протеазой. Очищенный слитой полипептид AnfK был сильно сконцентрирован во фракции элюата по сравнению с входящей фракцией, что демонстрируется интенсивностью полосы AnfK на мембране для блоттинга. При повторном анализе вестерн-блот с антителом HA, все кодированные слитые полипептиды Anf и Nif были обнаружены во входящей фракции, хотя полоса для AnfG стала видна только после экспозиции на мембране для блоттинга в течение 20 мин, а не 1 мин. Важно отметить, что антитело НА также показало присутствие процессированного полипептида AnfD в образце элюата. Присутствие AnfD и AnfK в образце элюата указывало на то, что после очистки слитого полипептида AnfK, MPP-процессированный полипептид AnfD очищали совместно, что указывает на белок-белковое взаимодействие этих двух слитых полипептидов.
Продуцирование гомоцитрата в инфильтрированных растительных клетках
Как описано в Примере 15, слитой полипептид NifV (AvNifV) кодон-оптимизированный в растении A. vinelandii, проявлял активность гомоцитрат-синтазы при индивидуальной экспрессии из генетической конструкции SN254. Образцы листьев, инфильтрированные либо SL42, либо SL43, либо обоими векторами в комбинации, анализировали на наличие гомоцитрата с использованием хроматографии GC-MS/MS, как описано в Примере 15. Гомоцитрат обнаружили в образцах, инфильтрированных SL43, либо отдельно, либо в сочетании с SL42, но не только с одним SL42. Это соответствовало наличию гена AvNifV в SL43.
Дальнейшие конструкции для комбинаций генов Anf и Nif
Как показано в Примере 20, митохондриально-нацеленный слитой полипептид, который соединил AnfD с AnfK с помощью олигопептидного линкера, экспрессировали, эффективно процессировали и наблюдали, его преимущественное присутствие во фракции растворимого белка после введения генетической конструкции в растительные клетки. Поэтому была создана генетическая конструкция, которая заменила гены AnfD и AnfK в SL43 гибридным геном, который кодировал слитой полипептид MTP-CoxIV::TwinStrep::AnfD::линкер26(HA)::AnfK (SEQ ID NO:203). Этот новый вектор получил обозначение SL48.
После ввода по отдельности SL48 и SL49 в листья N. benthamiana, с помощью анализа вестерн-блот наблюдали, что все кодированные полипептиды присутствовали, по меньшей мере, в некоторой степени в экстрактах растворимых белков (Рисунок 35 и 36). После ввода по отдельности SL48 и SL49 в листья N. benthamiana, с помощью анализа вестерн-блот наблюдали, что все восемь кодированных полипептидов присутствовали в экстрактах растворимых белков, включаяпроцессированный слитой полипептид scar::TwinStrep::AvAnfD::линкер26(HA)::AvAnfK(обозначенный как AnfDK на Рисунке 37).
Была изготовлена другая конструкция SL78 (Таблица 22), которая была аналогична SL49, за исключением добавления пятого гена, кодирующего MTP-FAγ51::NifS::HA. SL48 и SL78 инфильтрировали в листья N. benthamiana по отдельности или в комбинации. Анализ вестерн-блот фракций общих, растворимых и нерастворимых белков показал наличие всех кодированных слитых полипептидов в растворимой фракции, а также во фракции общего белка. То есть можно обнаружить все 9 слитых полипептидов, кодированных комбинацией из двух векторов, включая MPP-процессированный слитой полипептид scar9::TwinStrep::AvAnfD::Linker26(HA)::AvAnfK из SL48. Таким образом, все десять белков Anf и Nif, которые, как сообщается, необходимы в качестве минимального набора для образования Fe-нитрогеназы в бактерии E. coli (Yang et al., 2014), продуцировали в митохондриально-нацеленных растительных клетках и присутствовали, по меньшей мере, частично в растворимой форме.
Продуцирование гомоцитрата обнаруживается в инфильтрированных клетках, которые получили SL48.
Очистка белков из растительных клеток, продуцирующих слитые полипептиды Anf и Nif
Поскольку процессированный полипептид, кодированный геном MTP-CoxIV::TwinStrep::AnfD::линкер26(HA)::AnfK в SL48, имел эпитоп TwinStrep, трансляционно слитой после последовательности MTP на его N-конце, то для очистки этого слитого полипептида от клеток N. benthamiana, которые были инфильтрированы с SL48 и SL49, можно использовать колонку StrepTactinXT. Очистку проводили с использованием вышеописанных способов, а очищенный полипептид sscar::TwinStrep::AnfD::линкер26(HA)::AnfK концентрировали, используя способ, описанный в Примере 14.
Было замечено, что раствор, содержащий очищенный полипептид, был немного коричневого цвета у основания образца. Авторы изобретения посчитали, что этот цвет обусловлен присутствием кластеров Fe-S, связанных с полипептидом scar::TwinStrep::AnfD::линкер26(HA)::AnfK, что указывает на активность, по меньшей мере, слитых полипептидов NifS, NifU и AnfH в растительных клетках в предоставлении кластеров Fe-S слитому полипептиду AnfD-линкер-AnfK. Это подтверждается измерением содержания Fe2+ и S в выделенном полипептиде с использованием, например, масс-спектрометрии с индуктивно связанной плазмой (ICPMS). Ожидается, что измерения электропарамагнитного резонанса (EPR) позволят выявить определенные сдвиги длины волны, указывающие на присутствие и структуру кластеров Fe-S, связанных с полипептидом.
Увеличение числа связанных кластеров Fe-S ожидается за счет добавления другого гена к комбинациям генов Anf+Nif, описанным выше, а именно гена, кодирующего ферредоксин, например, FdxN из A. vinelandii или другого азотфиксирующего организма (Пример 22).
Пример 22. Экспрессия FdxN в растительных клетках с митохондриальным нацеливанием
Введение
Ген FdxN важен для оптимального функционирования нитрогеназы у многих диазотрофов, например у A. vinelandii (Jimenez-Vicente et al., 2014; Burén et al., 2019). Геном штамма CA A. vinelandii (Setubal et al., 2009; www.ncbi.nlm.nih.gov/nuccore/NC_021149.1) содержит 16 ферредоксиноподобных генов, включая FdxN, которые принадлежат к классу кластерных ферредоксинов 2x [4Fe- 4S] (Jimenez-Vicente et al., 2014).Данный класс ферредоксинов содержит два консервативных мотива, Cys-X2-Cys-X2-Cys-X3-Cys и Cys-X2-Cys-X7∼9-Cys-X3-Cys-X3∼5-Cys, которые консервируются в FdxN A. vinelandii за исключением последнего остатка Cys во втором мотиве (Matsubara и Saeki, 1992). Гены FdxN, функционирующие для нитрогеназы у бактерий, часто, но не всегда, обнаруживаются как часть оперона, транскрибируемого с другими генами, участвующими в нитрогеназе, включая гены Nif.. Например, FdxN в A. vinelandii является частью единственного оперона, содержащего NifB, FdxN, NifO-NifQ, RhdN и кодирующие белки области Grx5nif. FdxN транскрибировали примерно на том же уровне, что и NifB, в условиях диазотрофного роста. (Rodriguez-Quinones et al., 1993). Когда ферменты нитрогеназы экспрессировали в мутанте с делецией ΔFdxN из A. vinelandii, то наблюдали 5-кратное снижение синтеза NifB-ко и, следовательно, активности нитрогеназы. Таким образом, ген FdxN из A. vinelandii кодирует белок ферредоксин, который участвует в синтезе NifB-ко, что необходимо для всех трех Mo-, V- и Fe-нитрогеназ. Делеция FdxN также снижает скорость роста A. vinelandii в диазотрофных условиях до примерно 50% от дикого типа, указывая на то, что полное отсутствие FdxN допустимо для роста и активности нитрогеназы, но необходимо для оптимального роста и активности нитрогеназы. Считается, что FdxN в A. vinelandii действует либо как ферредоксин, отдавая электроны белку NifB во время продуцирования NifB-ко, либо как промежуточный переносчик [4Fe-4S] в NifB, либо и то, и другое (Burén et al., 2019 ).
Напротив, было продемонстрировано, что FdxN в Rhizobium meliloti необходим для симбиотической фиксации азота, поскольку мутанты fdxN неспособны фиксировать азот. Функцию восстановили путем введения плазмиды, кодирующей FdxN (Klipp et al., 1988). Очищенный полипептид FdxN R. meliloti был способен опосредовать перенос электронов к нитрогеназе Rhodobacter capsulatus in vitro (Riedel et al., 1995). Однако эта абсолютная потребность в FdxN у R. meliloti не отражалась на многих других диазотрофах, например, R. capsulatus.
Опять же, у K. oxytoca флаводоксин (NifF) и пируват:флаводоксин-оксидоредуктаза (NifJ) опосредуют перенос электронов от пирувата к нитрогеназе, а не к FdxN (Shah et al., 1983). В соответствии с этим кластер генов Nif K. oxytoca, продуцирующий функциональную нитрогеназу при переносе в E. coli, имел гены NifJHDKTYENXUSVWZMFLABQ, но не включал FdxN или эквивалентный ген (Smanski et al., 2014; Yang et al., 2013; Temme et al., 2012). Синтетический вектор pMIT v2.1 экспрессировал функциональную нитрогеназу в E. coli без включения гена FdxN, хотя и эндогенные ферредоксины в E. coli могли обеспечивать такую функцию. Белки, отличные от ферредоксина, также могли замещать функцию FdxN в E. coli, например флаводоксины. Нитрогеназа в диазотрофных бактериях обычно использует один или несколько белков флаводоксина, например, NifF и NifJ, в качестве донора электронов, поэтому NifF мог обеспечивать эту функцию. В другом исследовании, Yang et al. (2017), NifF K. oxytoca из вектора нитрогеназы pKU7017 заменили на Chlamydomonas или ферредоксины пластиды растения из Arabidopsis, кукурузы, риса и кукурузы, все из которых снижали уровень ацетилена на 50-100% по сравнению с контролем с NifF, показывая, что данные ферредоксины могут заменять NifF, по меньшей мере, в функции донорства электронов к белкам нитрогеназы NifH и NifD-NifK. Вектор pKU7017 не включает ген ферредоксина K. oxytoca, но имеет ген NifF, поэтому белок NifF или эндогенный ферредоксин E. coli могли обеспечивать электронами NifH/NifD-NifK или NifB для образования NifB-ко, или и того и другого. Напротив, Yates в 1972 обнаружил, что очищенный флаводоксин A. chromococcum, но не ферредоксин, может отдавать электроны зрелой динитрогеназе. В работе Jimenez-Vincente et al. (2014) подтвердили отсутствие передачи электронов FdxN в NifD-NifK. Следовательно, функция белка FdxN и его потребность для функции нитрогеназы не ясны для различных бактерий, не говоря уже о нитрогеназе, в ходе ее экспрессии в растениях и нацеливании в митохондрии.
Структура и разнообразие ферредоксинов и родственных белков были рассмотрены в работе Matsubara и Saeki (1992).
Филогенетический анализ полипептидов FdxN
Поиск в базе данных NCBI неизбыточных белков с использованием запроса FdxN A. vinelandii (SEQ ID NO:232) показал совпадение с семейством белков PRK13795 (гипотетический белок, предварительно), которое было единственным членом суперсемейства cl36298. Однако 627 аминокислотных последовательностей в PRK13795 кодировали ферменты, связанные с фосфоаденозин-фосфосульфат-редуктазой, обнаруженные у архей, которые имели в длину 400-800 аминокислот и содержали сайт связывания [4Fe-4S], но не содержали ферредоксин-подобных белков. Информация о белке для FdxN из штамма DJ A. vinelandii (Учетный №. ACO81189.1) и штамма CA (WP_012703542.1) аннотировала принадлежность к семейству pfam12838.. Область этого домена носила название «дикластерный домен Fer4_7 4Fe-4S», а pfam12838 был единственным членом семейства суперсемейства cl38378. Описание pfam12838 гласило: «Суперсемейство включает белки, содержащие домены, которые связываются с железо-серными кластерами». Члены семейства включают бактериальные ферредоксины, различные дегидрогеназы и различные редуктазы. Структура домена представляла собой альфа-бета-сэндвич, а домен содержал два кластера Fe4S4. В семействе белков pfam12838 было перечислено 206 репрезентативных аминокислотных последовательностей, из которых 26 аминокислотных последовательностей были короче 160аа, что использовалось в качестве ограничения размера, поскольку самая длинная последовательность из 16 последовательностей A. vinelandii, аннотированных как ферредоксин, составляла 156 остатков. Выравнивание 26 аминокислотных последовательностей в pfam12838 длиной 93-156 аминокислот осуществляли с помощью NCBI Global alignment (blast.ncbi.nlm.nih.gov/Blast) и определяли процент идентичности с SEQ ID NO:232 (WP_012703542.1). Процент идентичности 26 последовательностей относительно SEQ ID NO:232 составлял от 10 до 22%, что свидетельствует о разнообразии последовательностей FdxN. Использованные в этом анализе 26 последовательностей имели Учетный №.: Q8KG02_CHLTE, Q3ATN2_CHLCH, Q8KG03_CHLTE, Q9X2D5_THEMA, Q2JP81_SYNJB, Q9I1H8_PSEAE, Q01ZR2_SOLUS, ESU39497, WP_043013856, WP_012106131, WP_018723072, EKY12520, WP_012422852, ABG77170.1, EEX22670, WP_015853105, WP_012455913, WP_020095796, WP_012235387, WP_011973256, WP_015758977, WP_012302957, WP_012301895, WP_036081271, WP_004845399 и Q39V82_GEOMG.
Конструкции единичного гена для экспрессии слитых полипептидов FdxN в растительных клетках
Авторы изобретения стремились экспрессировать ген, кодирующий слитой полипептид FdxN A. vinelandii, в растительных клетках следующим образом, путем митохондриальной локализации продукта гена FdxN. Сначала исследовали две генетические конструкции (SN291, SN292), которые сами по себе экспрессировали слитой полипептид MTP-FdxN. В последующих экспериментах ген FdxN объединили в 5-генную конструкцию вместе с генами, кодирующими слитые полипептиды AnfD-линкер (HA) -AnfK, AnfH, AnfG и NifV, т. е. с 5 генами в одном векторе T-DNA и каждым слитым полипептидом, имеющим последовательность MTP для митохондриального нацеливания. Дальнейший эксперимент представлял собой совместную экспрессию с двумя конструкциями из 5 генов, а именно с вектором, кодирующим AnfD-линкер(HA)-AnfK, AnfH, AnfG, NifV и слитые полипептиды FdxN в одном векторе T-DNA, обозначенными SL50 и SL49 (Пример 21).
Две генетические конструкции SN291 и SN292 были сконструированы и изготовлены для экспрессии слитых полипептидов FdxN (SEQ ID NO:233, SEQ ID NO:234) отдельно в растительных клетках, например, клетках листьев N. benthamiana, а также были сконструированы и изготовлены две контрольные конструкции SN299 и SN300 (Таблица 23). Каждый синтетический ген находился под контролем сильного промотора 35S CaMV и области nos 3’ полиаденилирования/терминатора транскрипции, которые фланкировали кодирующую белок область. Аминокислотную последовательность FdxN из A. vinelandii (SEQ ID NO:232) с добавленным остатком Ala на С-конце использовали для создания в каждой конструкции нуклеотидной последовательности кодирующей белок области с кодон-оптимизацией, для экспрессии в растительных клетках. Для митохондриальной локализации SN291 кодировала слитой полипептид, содержащий последовательность MTP-FAγ51 слитую с N-концевым и С-концевым эпитопом НА для обнаружения полипептида в ходе анализа вестерн-блот с использованием антитела анти-НА. Эпитоп НА был слит трансляционно либо на С-конце (SN291), либо между MTP и последовательностью FdxN (SN292). Одна контрольная конструкция (SN300) кодировала полипептид, у которого отсутствовала последовательность MTP и, следовательно, меньше экспрессировала полипептид нацеленный на цитоплазму, который обеспечивал сравнение молекулярной массы в ходе анализа вестерн-блот для MPP-процессированного полипептида из полипептидов MTP-FdxN (процессированного FdxN), с подтверждением того, что MPP-процессированный полипептид в каждом случае включал «последовательность scar» примерно из 9 аминокислот, и поэтому размеры были близки но не идентичны. Вторая контрольная конструкция (SN299) кодировала слитой полипептид, который имел 13 аминокислот в последовательности MTP, замещенных аланинами (Allen et al., 2017), предназначенных для предотвращения MPP-процессинга. Таким образом, эти вторые контрольные полипептиды обеспечивали сравнение молекулярной массы для непроцессированного полипептида из соответствующих конструкций MTP-FdxN. Последовательность MTP с мутацией аланина была обозначена mFAγ51. После анализа белковых экстрактов из инфильтрированных тканей растения, образец из каждой конструкции MTP-FdxN и двух его соответствующих контрольных конструкций загружали на соседние дорожки для гель-электрофореза, что позволило наилучшим образом обнаруживать процессинг полипептида MTP-FdxN.
Таблица 23. Одиночная и мультгенная генетическая конструкция, кодирующая FdxN и компоненты Fe-нитрогеназы для экспрессии в растительных клетках
линкер26(HA)::AnfK
Продуцирование слитых полипептидов в растительных клетках
Культуры A. tumefaciens, содержащие SN291, инфильтрировали в листья N. Benthamiana возрастом 5-недель, как описано в Примере 1. Образцы листьев собирали через четыре-пять дней после инфильтрирования. Фракции общих, растворимых и нерастворимых белков экстрагировали следующим образом. Для исследования растворимости полипептидов, экспрессированных растением, ткань листа растирали в охлажденном на льду связывающем буфере для экстракции (100 ммоль Трис pH 8,0, 150 ммоль NaCl, 0,25 моль/л маннита, 5% (об./ об.) глицерина, 1% (об. об.) Tween® 20, 1% (мас./об.) PVP, только что добавленные 2 ммоль ТСЕР, 0,2 ммоль PMSF и 10 мкмоль/л леупептина) и переносили в микроцентрифужную пробирку. Образец центрифугировали при 20000 x g в течение 5 минут для разделения образца на растворимую (надосадочную) и нерастворимую (осадочную) фракции. Надосадочную жидкость переносили в чистую микроцентрифужную пробирку и снова центрифугировали при 20000 x g в течение 5 минут, для удаления любого оставшегося нерастворенного вещества. Нерастворимую фракцию промывали путем ресуспендирования осадка в 300 мкл буфера для экстракции с диспергированием повторными движениями пипетки и центрифугировали при 20000 x g в течение 5 мин, удаляя надосадочную жидкость. Эту стадию промывки повторяли еще дважды, удаляя любой оставшийся растворимый белок из нерастворимой фракции. Образцы затем подвергали электрофорезу SDS-PAGE и анализу вестерн-блот с использованием антитела анти-HA. Антитело анти-НА (моноклональное анти-HA, Sigma) использовали разведенным в соотношении 1:5000.
Анализ вестерн-блот для SN291 (Рисунок 38) показал, что полипептид FdxN легко обнаруживался во фракции общего белка с HA-антителом, показывая слабую интенсивность полипептида, присутствующего как в растворимой, так и в нерастворимой белковой фракциях, что требует более длительных экспозиций в ходе анализа вестерн-блот для улучшения видимости, что также указывает на частичную растворимость слитого полипептида AvFdxN. Слитой полипептид FdxN, по-видимому, частично процессируется с помощью MPP как в процессированных, так и в непроцессированных формах, что указывает на неэффективное расщепление с помощью MPP. Полосы на соседних дорожках для контрольных полипептидов в каждом случае подтверждали, что эти полосы были для процессированных и для непроцессированных полипептидов.
Культуры A. tumefaciens, содержащие SN292, инфильтрировали в листья N. Benthamiana возрастом 5-недель, как описано в Примере 1. Образцы листьев собирали через четыре-пять дней после инфильтрирования. Фракции общих, растворимых и нерастворимых белков экстрагировали, используя тот же способ, что и для SN291. Анализ вестерн-блот для SN292 показал, что полипептид FdxN легко обнаруживается во фракции общего белка с антителом HA, что указывает на то, что позиция эпитопа HA в слитом полипептиде либо С-конце, либо по направлению к N-концу, не влияет на уровень экспрессии полипептида. Опять же, по-видимому, слитой полипептид FdxN частично процессируется MPP и при этом большая часть белка имеет правильный размер для процессированных форм. Полосы на соседних дорожках для контрольных полипептидов в каждом случае подтверждали, что эти полосы были для процессированных полипептидов.
Продуцирование комбинаций слитых полипептидов, включающих FdxN, в растительных клетках
Новая генетическая конструкция была разработана и изготовлена методами сборки GoldenGate, обозначена SL50 (Таблица 22) и исследована отдельно (Рисунок 40) и в комбинации с SL49 или SL54 (Рисунок 41). Один ген в SL50 кодировал слитой полипептид MTP-CoxIV::TwinStrep::AnfD::линкер26(HA)::AnfK, а другие четыре гена кодировали слитые полипептиды AnfH, AnfG, NifV и FdxN, каждый из которых имел последовательность MTP-FAγ51 с последующим трансляционным слиянием эпитопа НА с N-концом полипептидов. Генетические конструкции SL49 и SL50 вводили в клетки N. benthamiana по отдельности, а экспрессию белка анализировали в ходе анализа вестерн-блот. Все пять кодируемых слитых полипептидов из SL50 обнаруживали с помощью соответствующих антител, каждое из которых показывало полосы полипептида, присутствующие во фракции растворимого белка, за исключением полипептида FdxN, который не был виден ни в растворимой, ни в нерастворимой фракциях (Рисунок 40). Важно отметить, что процессированные слитые полипептиды AnfD-линкер-AnfK, NifV и AnfH наблюдали как в растворимой фракции, так и в нерастворимой фракции, следовательно все три были, по меньшей мере, частично растворимы. Полипептиды AnfG, AnfH и NifV, по-видимому, частично расщеплялись, поскольку каждый из них показывает полосы для процессированных и непроцессированных форм. Полипептид AnfD-линкер-AnfK, по-видимому, эффективно процессировался. Полипептид FdxN был виден только после длительной экспозиции и только при процессированном размере в пределах фракции общего белка.
Затем культуры Agrobacterium, содержащие SL50 и SL49, смешивали и инфильтрировали в листья N. benthamiana, как описано ранее. Таким образом, в этом эксперименте вводили: 3 гена Anf, кодирующие полипептиды AnfH и AnfG, и слитой полипептид AnfD-линкер-AnfK; 5 генов Nif, кодирующих полипептиды NifF, NifJ, NifU, NifB и NifV, и ген FdxN, т.е. 9 генов в комбинации. Неожиданный результат, наблюдавшийся в ходе анализа вестерн-блот (Рисунок 41), заключался в том, что все 9 полипептидов легко обнаруживались. Некоторые полипептиды были видны в виде 2 полос, причем верхняя полоса представляла непроцессированный полипептид, а нижняя полоса представляла расщепленный полипептид при импорте в митохондрии. Полоса непроцессированного полипептида была видна для слитых полипептидов NifV, NifU и NifF, как и полоса предсказанного размера для расщепленных полипептидов. Все полипептиды, за исключением полипептида FdxN, присутствовали в растворимой фракции, которая не была видна ни в растворимой, ни в нерастворимой фракциях из-за низкого уровня накопления.
Другая генетическая конструкция была разработана и изготовлена методами сборки Golden Gate, обозначена SL54 (Таблица 23) и исследована отдельно и в комбинации с SL50. SL54 имел ген, кодирующий слитой полипептид MTP-FAγ51::NifB::HA (SEQ ID NO:147), в котором использовали последовательность из K. oxytoca, но в остальном SL54 был идентичен SL42 (Таблица 22) для экспрессии слитых полипептидов NifS, NifU, NifJ и NifF. В этом эксперименте также исследовали, может ли слитой полипептид NifB, на основе последовательности K. oxytoca, которая ранее показала наибольшую нерастворимость при отдельной экспрессии, улучшить растворимость при экспрессии в комбинации с другими полипептидами.
SL50 и SL54 сначала вводили отдельно в листья N. benthamiana, а фракции растворимых и нерастворимых белков получали и анализировали с помощью вестерн-блот, как и фракции общего белка. Было обнаружено, что все кодированные полипептиды присутствуют в экстрактах растворимых белков, по меньшей мере, в некоторой степени, а также во всех фракциях общего белка, за исключением полипептидов NifB и FdxN, которые были либо не видны, либо затемнены другими полосами белка подобного размера. Полипептид с наименьшей интенсивностью представлял собой полипептид FdxN, который был виден только в образце общего белка и только после более длительной экспозиции.
SL50 и SL54 также вводили вместе в листья N. benthamiana. По данным анализа вестерн-блот полипептиды AnfH, AnfG, NifV, NifJ, NifS, NifU и NifF, а также процессированный полипептид AnfD-линкер-AnfK, по меньшей мере, в некоторой степени присутствуют в экстрактах растворимых белков. Опять же, полипептид FdxN не был виден ни в растворимой, ни в нерастворимой фракциях из-за низкого уровня накопления. Присутствие полипептида NifB в растворимой фракции не могло быть подтверждено из-за его размера, который совпадал с размером непроцессированного полипептида NifS при электрофорезе в полиакриламидном геле в присутствии додецилсульфата натрия (Рисунок 39).
В качестве контроля размера и растворимости вектор (SN192) с одним геном, кодирующий полипептид NifB K. oxytoca, отдельно инфильтрировали в листья N. benthamiana. При анализе вестерн-блот с антителом HA, полипептид NifB был виден как в непроцессированной, так и в процессированной формах и был виден во фракции общего белка и нерастворимой фракции, при этом NifB не было видно в растворимой фракции (Рисунок 39).
В дальнейших экспериментах будут очищать полипептид TwinStrep-AnfD-линкер-AnfK, экспрессированный из SL50. В дальнейших экспериментах будут исследованы комбинации SL50 с вариантами SL54, причем с вариантами имеющими полипептиды NifB, происходящие от организмов, отличных от K. oxytoca.
Получение растений, стабильно трансформированных генами Anf и Nif
Набор генов в каждом из SL49 и SL50 переносили отдельно в бинарный вектор, имеющий селектируемый маркерный ген. Полученные конструкции использовали для создания трансформированных растений A. thaliana. После первоначального отбора с использованием соответствующего антибиотика девять трансформантов Т1 получали для SL49 и два трансформанта Т1 получали для SL50. Конструкции также использовали для трансформации табака (N. tabacum) и N. benthamiana. Ожидается, что эти трансгенные растения будут экспрессировать все кодированные полипептиды, включая кластеры Fe-S, например, P-кластеры в полипептидах AnfD-линкер-AnfK и AnfH, и продуцировать гомоцитрат в больших количествах, по сравнению с соответствующими растениями дикого типа или растениями, лишенными гена NifV. Также ожидается, что растения будут иметь позитивную регуляцию для продуцирования кластеров Fe-S, например Р-кластера в полипептидах AnfD-линкер-AnfK и AnfH, и будут продуцировать гомоцитрат в больших количествах, по сравнению с растениями дикого типа. Растения исследуются на активность Fe-нитрогеназы.
Пример 23. Анализ полипептидов Anf
В контексте данного документа «полипептид AnfH» представляет собой полипептид NifH, который является элементом консервативного суперсемейства cl25403 нитрогеназы, содержащего консервативный домен PRK13233, и который, по меньшей мере, идентичен на 69% аминокислотной последовательности полипептида AnfH Azotobacter vinelandii (SEQ ID NO:218) при измерении полноразмерной SEQ ID NO:218 из 275 аминокислотных остатков. Авторы изобретения проанализировали полипептидные последовательности AnfH, присутствующие в базах данных, выровняли их и сравнили с репрезентативным NifH молибденового типа.
В базах данных провели поиск аминокислотных последовательностей AnfH. Было обнаружено, что они имеют консервативный домен PRK13233, который, по меньшей мере, идентичен на 69% SEQ ID NO:218. Было обнаружено 314 таких последовательностей. Их выровняли с помощью инструмента NCBI COBALT и разработали обобщающую типичную последовательность, которая имела 300 позиций остатков, включая пробелы. Эта обобщающая типичная последовательность была идентична на 89% SEQ ID NO:218. Удивительно то, что выровненные аминокислотные последовательности AnfH содержали 137 аминокислот из 300 позиций, которые были идентичны для всех из 314 встречающихся в природе полипептидов AnfH и для многих других аминокислот, консервативных во многих полипептидах AnfH. Поскольку 137 консервативных аминокислот в домене PRK13233 охватывали большую часть последовательностей AnfH, то был сделан вывод, что домен PRK13233 охватывал большую часть последовательностей AnfH и что PRK13233 указывал на семейство последовательностей, а не на одну конкретную последовательность. Указанные 137 консервативных аминокислот содержали мотивы последовательностей YGKGGIGKSTTXQNT (мотив I, SEQ ID NO:225), IHGCDPKAD (мотив II, SEQ ID NO:226), CVESGGPEPGVGCAGRG (мотив III, SEQ ID NO:227), DVLGDVVCGGFAMP (мотив IV, SEQ ID NO:228), VASGEMMAXYAANNI (мотив V, SEQ ID NO:229), QSGVR (мотив VI, SEQ ID NO:230) и CNSRXVD (мотив VII, SEQ ID NO:231), где X представляет собой любую аминокислоту. Все мотивы I-VII присутствовали во всех 314 проанализированных последовательностях AnfH.
Следующие 137 аминокислот, которые были полностью консервативными обозначены номером, относящимся к позиции аминокислоты в SEQ ID NO:218, и буквой аминокислоты в этой позиции: 3R, 4K, 6A, 8Y, 9G, 10K, 11G, 12G, 13I, 14G, 15K, 16S, 17T, 18T, 20Q, 21N, 22T, 25A, 36I, 37H, 38G, 39C, 40D, 41P, 42K, 43A, 44D, 46T, 47R, 50L, 52G, 55Q, 60D, 63R, 75V, 79G, 85C, 86V, 87E, 88S, 89G, 90G, 91P, 92E, 93P, 94G, 95V, 96G, 97C, 98A, 99G, 100R, 101G, 103I, 104T, 106I, 108L, 109M, 110E, 115Y, 119L, 120D, 125D, 126V, 127L, 128G, 129D, 130V, 131V, 132C, 133G, 134G, 135F, 136A, 137M, 138P, 140R, 142G, 143K, 144A, 146E, 148Y, 150V, 151A, 152S, 153G, 154E, 155M, 156M, 157A, 159Y, 160A, 161A, 162N, 163N, 164I, 167G, 170K, 172A, 174Q, 175S, 176G, 177V, 178R, 180G, 181G, 184C, 185N, 186S, 187R, 189V, 190D, 192E, 198E, 199F, 204G, 212P, 213R, 215N, 217V, 218Q, 220A, 221E, 222F, 227V, 236Q, 239E, 240Y, 243L, 247I, 250N, 254V, 255I, 256P, 258P, 265E, 272G. В ходе выравнивания с последовательностью NifH A. vinelandii (AvNifH; SEQ ID NO:224) 121 из 137 полностью консервативных аминокислот из последовательностей AnfH также присутствовала в соответствующих позициях AvNifH. Указанные 16 аминокислот, которые были консервативными во всех последовательностях AnfH, но не в AvNifH, представляли собой: 4K, 22T, 37H, 52G, 60D, 63R, 108L, 109M, 142G, 151A, 174Q, 189V, 198E, 199F, 222F и 247I со ссылкой на SEQ ID NO:218. Таким образом, эти 16 аминокислот были характерными для AnfH, по сравнению с последовательностью NifH молибденового типа из AvNifH, и могут использоваться для различения полипептидов AnfH от других последовательностей NifH, которые не имеют всех 16 общих аминокислот. AvNifH, KoNifH (SEQ ID NO:1) и другие последовательности NifH молибденового типа имели мотивы III и IV, но не имели мотивов I, II, V-VII, и поэтому эти мотивы также могли использоваться для отличия подмножества AnfH от других полипептидов NifH.
Пример 24. Совместная экспрессия дополнительных полипептидов Nif улучшает содержание комплекса NifD-NifK и NifY
Зрелая и каталитически активная Mo-нитрогеназа включает два металлофактора, P-кластер и кластер FeMo-ко. Эти металлокластеры собираются в несколько этапов в порядке, описанном в работе Burén et al. (2019), в значительной степени основанном на исследованиях нитрогеназы A. vinelandii. Для синтеза P-кластера полипептид NafH взаимодействует с белковым комплексом, названным пре-апо-NifD-NifK, и способствует размещению переданных из NifU 2-х отдельных кластеров [Fe4-S4] в позиции внутри полипептидов NifD и NifK. Затем взаимодействие NafH-NifD-NifK заменяли на взаимодействие NifW-NifD-NifK. Затем полипептид NifW замещается зрелыми NifH и NifZ, и на этой стадии кластеры [Fe4-S4] конденсируются на границе раздела NifD и NifK в кластер [Fe8-S7], так называемый P-кластер с элиминацией одного атома серы. Образование P-кластера превращает пре-апо-NifD-NifK в один, возможно, два промежуточных продукта «апо-NifD-NifK», которые связываются с NafY (также называемым γ-белком) и/или NifY. В случае с NafY структурные исследования показали, что N-концевой домен в NafY связывается с апо-NifD-NifK, а C-концевой домен связывается с FeMo-ко. FeMo-ко образуется где-то еще в NifE-NifN и считается, что NifX участвует в перемещении металлофактора между белками.
Этот путь последовательной сборки и его предполагаемые белковые взаимодействия основаны на исследованиях нитрогеназы A. vinelandii, и некоторые из этих этапов, вероятно, отличаются или используют разные белки в других организмах. Например, в Klebsiella oxytoca отсутствуют гены, кодирующие NafH или NafY, а в A. vinelandii его NifY больше похож на NafY, чем на NifY. NifX в K. oxytoca был не нужен для диазотрофии (Temme et al., 2012). Для образования P-кластеров в Klebsiella требуется только функциональный Fe-белок (NifH), поскольку делеция гена NifH нарушает формирование P-кластеров и диазотрофный рост. Напротив, отдельная делеция генов, кодирующих NafY, NifY, NifW или NifZ в A. vinelandii, замедляла, но не останавливала диазотрофный рост, указывая на то, что эти компоненты были частично избыточными или, что недостаток определенного белка мог быть компенсирован другими факторами в A . vinelandii.
Авторы настоящего изобретения решили исследовать влияние совместной экспрессии полипептидов NifW, NifX, NifY и NifZ в растительных клетках с митохондриальным нацеливанием на слитой полипептид NifD-NifK. Для этого была создана конструкция для экспрессии растений, обозначенная SL55, с использованием метода сборки Golden Gate. SL55 имел четыре гена Nif, кодирующих слитые полипептиды KoNifW, KoNifX, KoNifY и KoNifZ, каждый на основе последовательности K. oxytoca, имея N-концевое слияние с последовательностью MTP-FAγ51. Каждый полипептид также имел эпитоп НА, слитой на С-конце, для обнаружения при анализе вестерн-блот. Компоненты, использованные для создания SL55, были взяты из SN340 (MTP-KoNifW-HA), SN144 (MTP-KoNifX-HA), SN145 (MTP-KoNifY-HA) и SN146 (MTP-KoNifZ-HA). Каждый отдельный ген фланкировали 35S-промотором и областью 3’ последовательности полиаденилирования/терминатора транскрипции для экспрессии в растительных клетках. Второй генетической конструкцией, использованной в эксперименте по совместному инфильтрированию, была SL47, кодирующая митохондриально-нацеленный MTP-FAγ51::KoNifDY100Q::линкер26(HA)::KoNif, как кодированный SN159. Это трансляционное слияние имело последовательность NifD на основе последовательности K. oxytoca с заменой Y100Q в последовательности NifD. Конструкции SL55 и SL47 инфильтрировали либо отдельно, либо вместе в листья N. benthamiana, а через 4 или 5 дней после инфильтрирования отбирали образцы для анализа вестерн-блот. Белки экстрагировали в аэробных условиях, разделяли в 4-20% градиенте геля (SDS-PAGE) и анализировали с антителом анти-НА и вторичным антителом HRP, как и ранее.
Листья, инфильтрированные одной SL47, продуцировали относительно слабый сигнал для полипептида массой ~ 110 кДа, ожидавшийся для размера полипептида scar::NifD::линкер26(HA)::NifK (Рисунок 42). Листья, экспрессирующие четыре слитых гена MTP-Nif в SL55 либо отдельно, либо совместно инфильтрированные с SL47, продуцируют сильные сигналы для правильно MPP-процессированных полипептидов NifW, NifX, NifY и NifZ, как описано в Примерах 2 и 3. Неожиданным и существенным оказался тот факт, что листья, инфильтрированные совместно с SL55 и SL47, привели к гораздо большей интенсивности полосы, соответствующей правильно процессированному полипептиду scar::NifD::линкер26(HA)::NifK (Рисунок 42). Также было отмечено, что более слабая полоса, продуцированная SL55 при масссе примерно 100 кДа, возможно, возникавшая из-за вторичной деградации полипептида scar::NifD::линкер26(HA)::NifK в митохондриях, была менее обильной, когда SL47 была совместно инфильтрирована с SL55. Это уменьшенное содержание предполагаемого продукта деградации имело место, несмотря на большее содержание правильно процессированного полипептида scar::NifD::линкер26(HA)::NifK.
Кроме того, совместная экспрессия комбинации векторов SN340, SN144, SN145 и SN146 с одним геном приводила к большей интенсивности полосы для правильно процессированного NifY по сравнению с экспрессией только из SN145. Этот результат предполагает, что комбинация SN340, SN144 и SN146 (слитые полипептиды NifW, NifX и NifZ) улучшает экспрессию и/или стабильность слитого полипептида NifY в митохондриях растения. Авторы изобретения пришли к выводу, что один или более чем один, или комбинация митохондриально-нацеленных NifW, NifX, NifY и NifZ увеличивают содержание трансляционно слитых полипептидов NifD и NifK. Этот эксперимент также показал, что совместная экспрессия полипептидов NifW, NifX и NifZ увеличивает содержание NifY в растительных клетках.
Была изготовлена другая конструкция (SN229), кодирующая аналогичный слитой полипептид NifD-NifK, но включающая эпитоп Twin-strep для очистки MPP-процессированного полипептида из растительных клеток. SN229 совместно с SL55 была инфильтрирована в листья N. benthamiana. Белковые экстракты готовили и пропускали через колонку Strep-tactinXR в аэробных или анаэробных условиях. Элюат из колонки содержит очищенный полипептид scar::TS::NifD::linker26(HA)::NifK и анализируется на наличие слитого полипептида NifW и NifZ, один или оба из которых, как ожидается, будут очищаться совместно с белком NifD-NifK.
Конструкции SN299, SL55 и третья конструкция, кодирующая отдельные слитые полипептиды NifH, NifM, NifS и NifU, все митохондриально-нацеленные путем слияния с MTP, совместно инфильтрировали в листья N. benthamiana. Белковые экстракты вновь готовили и пропускали через колонку Strep-tactinXR в аэробных или анаэробных условиях. Ожидается, что полученный элюат будет содержать очищенный полипептид scar::TS::NifD::линкер26(HA)::NifK, который имеет правильно сформированный и связанный с ним P-кластер, способный аккумулировать FeMo-ко, т. е. полипептид апо-NifD-NifK. Уровень содержания P-кластера измеряется с помощью ICP-MS.
Настоящая заявка испрашивает приоритет согласно предварительной заявки на патент AU 2019900780, поданной 8 марта 2019 г., патент AU 2019903818 поданной 10 октября 2019 г., и патент AU 2020900689 поданной 5 марта 2020 г., содержание которых полностью включено в настоящую заявку посредством ссылки.
Специалистам в данной области техники очевидно, что могут быть сделаны различные модификации и вариации вышеописанного изобретения, без отклонения от сущности и объема настоящего изобретения. Настоящие варианты осуществления изобретения следует рассматривать во всех отношениях как иллюстративные и не ограничивающее объем изобретения.
Все публикации или ссылки на них полностью включены в настоящую заявку посредством ссылки.
Любое рассмотрение документов, фактов, материалов, устройств, изделий и т.п., включенное в настоящее описание, предназначено исключительно для конкретных условий настоящего изобретения. Это не следует принимать за признание, что любые или все эти вопросы являются частью базы данных предшествующего уровня техники или являются общеизвестными сведениями в области, относящейся к настоящему изобретению, как это существовало до даты приоритета каждого пункта данной заявки.
СПИСОК СПРАВОЧНОЙ ЛИТЕРАТУРЫ
Abe et al. (2000). Cell 100:551-560.
Abdullah et al. (1986). Biotechnology 4(12):1087.
Allen et al. (1994). Crit. Rev. Biotechnol. 14:225-249.
Allen et al. (1995). J. Biol. Chem. 270:26890-26896.
Allen et al. (2017). Front. Plant Sci., 8:287. doi: 10.3389/fpls.2017.00287
Arnold et al. (1988). Tree physiology, 4:291-300.
Altschul et al. (1997). Nucleic Acids Res. 25:3389-3402.
Balk and Pilon (2011). Trends Plant Sci 16:218-226.
Barker et al. (1983). Plant Molecular Biology, 2(6):335-350.
Becker et al. (2012). Trends in Biochemical Sciences 37:85-91.
Bevan et al. (1983). Nature 304:184-187.
Boison et al. (2006). Arch. Microbiol. 186:367-376.
Boyd et al. (2011) Geobiology 9:221-232
Boyd and Peters (2013). Front.Microbiol. 4:201. doi:10.3389/fmicb.2013.00201.
Brigle et al. (1987). J. Bacteriol. 169:1547-1553.
Bruce (2001). Biochimica et Biophysica Acta (BBA) - Molecular Cell Research 1541:2-21.
Burén et al. (2017a). Front Plant Sci 8:1567.
Burén et al. (2017b). ACS Synthetic Biology 6(6):1043-1055.
Burén and Rubio (2018) Fems Microbiol Lett 365:fnx274; doi: 10.1093/femsle/fnx274.
Burén et al. (2019). Proc. Nat. Acad. Sci. USA 116:25078-25086.
Burén et al. (2020). Chemical Reviews doi.org/10.1021/acs.chemrev.9b00489.
Cannon et al. (1988) Nucleic Acids Res. 16:11379.
Capecchi. (1980). Cell, 22(2): 479-488.
Carrari et al. (2003). Metab. Eng. 5:191-200.
Carrie et al. (2010). Journal of Biological Chemistry 285:36138-36148.
Carter et al. (1980). J. Biol. Chem. 255:4213-4223.
Chacinska et al. (2009). Cell 138:628-644.
Chen et al. (2013). Advanced Drug Delivery Reviews 65:1357-1369.
Cheng et al. (1996). Proceedings of the National Academy of Sciences, 93(8):3636-3641.
Cheng et al. (2005). Biochemical and Biophysical Research Communications 329:966-975.
Chiu et al. (2001). Biochemistry 40:641-650.
Christiansen et al. (1998) Biochemistry-Us 37: 12611-12623.
Clapp (1993). Clinics in perinatology, 20(1):155-168.
Clausen et al. (2000). Proc. Natl. Acad. Sci. U.S.A. 97:3856-3861.
Cotton (2009). J. Am. Chem. Soc. 131: 4558-4559.
Cui et al. (2013). Proceedings of the National Academy of Sciences 110, 2052-2057.
Curatti et al. (2006). Proc. Natl. Acad. Sci. U.S.A. 103:5297-5301.
Curatti et al. (2007). Proceedings of the National Academy of Sciences 104(45): 17626-17631.
Curatti and Rubio (2014). Plant Sci 225:130-137.
Curiel et al. (1992). Am J Respir Cell Mol Biol, 6(3): 247-252.
Darshi et al. (2012). Journal of Biological Chemistry 287:39480-39491.
Davis et al. (1996). J. Bacteriol. 178:1445-1450.
De'ath et al. (2012). Proc. Natl. Acad. Sci. USA 109:17995-17999.
de Bruijn (2015). In: Biological Nitrogen Fixation pp. 1087-1101. John Wiley & Sons, Inc.
de Kok et al. (2014). ACS Synth. Biol. 3:97-106.
Dilworth et al. (1988). Biochem. J. 249:745-751.
Dilworth et al. (1993). Biochem. J. 289:395-400.
Dos Santos et al. (2004). Chem Rev. 104:1159-73.
Dos Santos et al. (2012). BMC Genomics 13:162.
Drummond (1985). Biochem. J. 232:891-896.
Dyer et al. (2003). J. Biol. Chem. 278:32150-32156.
Eady (1996). Chem. Rev. 96:3013-3030.
Eglitis et al. (1988). Advances in Experimental Medicine and Biology 241:19-27.
Emerich and Burris, (1978). J. Bacteriol. 134:936-943.
Engler et al. (2014) ACS Synthetic Biology 3(11):839-843.
Fani et al. (2000). J. Mol. Evol. 51:1-11.
Fay et al. (2015) Proc Natl Acad Sci U S A 112: 14829-14833.
Fay et al. (2016). Proc. Natl. Acad. Sci. U.S.A. 2016:9504-9508.
Fujimura et al. (1985). Plant Tissue Cult Lett. 2:74-75.
Fukusawa et al. (2015). Molecular and Cellular Proteomics 14:10.1074/mcp.M114.043083, 1113-1126.
Gallie et al. (1987). Nucleic Acids Res. 15:3257-73.
Garfinkel et al. (1983). Cell 27:143-153.
Gavini et al. (1998). Biochemical and Biophysical Research Communications. 244:498-504.
Gavini et al. (2006). Journal of Bacteriology 188:6020-6025.
Geddes et al. (2015). Curr Opin Biotech 32:216-222.
Geigenberger and Fernie (2014). Antioxid Redox Sign 21:1389-1421.
Glaser and Deshi (1999). J Bioenerg Biomembr 31:259-274.
Glibert et al. (2014). Environ Res Lett 9:e105001; doi.org/10.1088/1748-9326/9/10/105001.
Glick et al. (1992). Cell 69:809-822.
Goodwin et al. (1998) Biochemistry-Us 37: 10420-10428.
Good and Beatty (2011). PLoS Biol 9, e1001124.
Graham et al. (1973). Virology 52(2) 456-467.
Grant et al. (1995). J. Agric. Sci., 124 (3): 437-445
Guo et al. (2016). Angewandete Chemie 55:12764-12767
Fu et al. (1994). Biochemistry 1994 33:13455-63.
Hakoyama et al. (2009). Nature 462(7272): 514.
Hellinga (1997). Proc Natl Acad Sci U S A. 94:19 10015-10017.
Hernandez et al. (2007). Mol. Microbiol. 63:177-192.
Hirel et al. (1989). Proc. Natl. Acad. Sci. USA 86:8247-8251.
Hinchee et al. (1988).
Homer et al. (1993). J. Bacteriol. 175:4907-4910.
Homer et al. (1995). J. Biol. Chem. 270:24745-24752.
Hoover et al. (1988). Biochemistry 27: 3647-3652.
Horsch et al. (1985). Cold Spring Harbor Symposia on Quantitative Biology 50:433-437.
Howard et al. (1986). J Biol. Chem. 261:772-778
J. Biol. Chem. 279:54963-54971. http://europepmc.org/abstract/MED/15485884
Hu et al. (2005). Proc. Natl. Acad. Sci. U.S.A. 102:3236-3241.
Hu et al. (2006). Proc. Natl. Acad. Sci. U.S.A. 103:17119-17124.
Hu et al. (2008). Biochemistry 47:3973-3981.
Hu and Ribbe (2013). Bba-Bioenergetics 1827:1112-1122.
Hu and Ribbe, (2015). Journal of Biological Inorganic Chemistry 20(2):435-445. doi:10.1007/s00775-014-1225-3
Hu and Ribbe (2016). Annual Review of Biochemistry 85:455-483.
Huang et al. (2009). Plant Physiology 150(3):1272-1285.
Hummel et al. (2007). Metabolomics 75-95.
Hwang et al. (1996). J. Mol. Evol. Nov; 43:536-540.
Igarashi and Seefeldt (2003). Crit. Rev. Biochem. Mol. Biol. 38:351-384.
Jasniewski et al. (2018). Inorganics 6(1):25.
Jiménez-Vicente et al. (2014) FEBS Letters 588:512-516.
Johnson et al. (2005). Biochem. Soc. Trans. 33:90-93.
Joshi (1987). Nucleic Acids Res. 15:6643-6653.
Jouanneau et al. (1995). Biochim. Biophys. Acta 1232:33-42.
Katoh et al. (2013). Mol Biol Evol. 4:772-80.
Kay et al. (1987). Science 236:1299-1302.
Kennedy and Dean, (1992). Mol Gen Genet. 231:494-498.
Kerscher et al. (1997). The Journal of Cell Biology 139:1663-1675.
Khumanthem et al. (2007). Indian Journal of Microbiology, 47:345-352.
Kim and Rees (1994). Biochemistry 33:389-397.
Kimble et al. (1995). Archives of Microbiology 163:259-267.
Klipp et al. (1988). Mol. Gen. Genet. 216:293-302.
Kmiec et al. (2013). PNAS 110: 40 E3761-E3769.
Koon et al. (2004). Proc Natl Acad Sci USA 10:8295-8300.
Lawson and Smith (2002). Met Ions Biol Syst; 39:75-119.
Lee et al. (1998). Biochemical and Biophysical Research Communications 244: 2 498-504.
Lee et al. (2000). J. Bacteriol. 182:7088-7091.
Lee et al. (2012). Plant Cell 24:5037-5057.
Lei et al. (1999). Biochem Biophys Res Commun. 264:186-90.
Lill and Mühlenhoff (2008). Annual Review of Biochemistry 77:669-700.
Lister et al. (2004). 134:777-789.
Lister et al. (2007). Plant Cell, 19:3739-3759.
Lopez-Torrejon et al. (2016). Nature Communications 7:11426.
Lu et al. (1993). Sci China B. 36: 11 1342-51.
Mackenzie and McIntosh (1999). Plant Cell 11:571-585.
Marques et al. (2014). Acta Crystallographica Section F 70(5):669-672.
Masukawa et al. (2007). Appl. Environ. Microbiol. 73:7562-7570.
Matsubara and Saeki (1992). Adv. Inorganic Chem. 38:223-280.
Mayer et al. (1999). J. Mol. Biol. 292:871-891.
McLean and Dixon (1981). Nature, 292:655.
McRose et al. (2017). Frontiers Microbiol. 8:267 doi:10.3389/fmicb.2017.00267.
Medberry et al. (1992). The Plant Cell 4: 2 185-192.
Medberry et al. (1993). Plant J. 1993 3: 4 619-26.
Merrick and Dixon (1984). Trends Biotechnol 2:162-166.
Mehta и Baross (2006). Science 314:1783-1786.
Millar et al. (2007). Methoods Cell Biol 80:65-90.
Miller and Eady (1988). Biochem. J. 256:429-432.
Miyamoto et al. (1979). Applied and Environmental Microbiology, 37:454-458.
Mueller et al. (2012). Nature 490:254-257.
Mühlenhoff et al. (2003). EMBO J. 22:4815-4825.
Murcha et al. (2004). J Mol Biol 344:443-454.
Murcha et al. (2014). Bba-Gen Subjects 1840:1233-1245.
Niedz et al. (1995). Plant Cell Reports 14: 7 403-6.
Naim et al. (2012). PLoS One 7(12):e52717.
Oldroyd and Dixon (2014) Curr Opin Biotechnol 26:19-24.
Olson et al. (2000). Biochemistry; 39:16213-16219.
Ouzounis et al. (1994). Trends Biochem. Sci. 19:199-200.
Ow et al. (1986). Science 234:856-859.
Paul and Merrick (1987) Eur. J. Biochem. 170:259-265.
Paustian et al. (1990). Biochemistry 29:3515-3522.
Petrova et al. (2000). Biochem. Biophys. Res. Commun. 270:863-867.
Pfanner and Geissler (2001) Nat. Rev. Mol. Cell Biol. 2:339-349.
Prasad et al. (1992). Plant Molecular Biology 18(5):873-885.
Prasher et al. (1985). Trends in Genetics 11: 8 320-3.
Pratte et al. (2006). J. Bacteriol. 188:5806-5811.
Riedel et al. (1995). Eur. J. Biochem. 231:742-746.
Robson and Postgate (1980). Annual Review of Microbiology 34:183-207.
Robson et al. (1986). Nature, 322:388-390.
Robson et al. (1989). EMBO J. 8:1217-1224.
Rockstrom et al. (2009). Nature 461:472-475.
Rodriguez-Quinones et al. (1993). J. Bacteriol. 175:2926-2935.
Roise et al. (1986) The EMBO Journal 5:1327-1334.
Roise and Schatz (1988). J.Biol.Chem. 263:4509-4511.
Rubio et al. (2002). J. Biol. Chem. 277:14299-14305.
Rubio et al. (2004) J Biol Chem 279: 19739-19746.
Rubio and Ludden (2005). J. Bacteriol. 187:405-414.
Rubio and Ludden (2008). Annu Rev Microbiol 62:93-111.
Salomon et al. (1984). EMBO 3:1 141-146.
Santi et al. (2013). Ann Bot 111:743-767.
Schleiff and Soll (2000) Planta 211:449-456.
Schmidt and Skerra (2007). Nat. Protoc. 2:1528-1535
Schmidt et al. (2013). Protein Expr. Purif. 92:54-61.
Schmitz et al. (2001). FEMS Microbiol Lett. 195:97-102.
Schwarz et al. (2016). Nucleic acids research 44(8): e77-e77.
Seefeldt et al. (2009). Annu Rev Biochem 78:701-722.
Serrano et al. (2009). Systematic and Applied Microbiology, 32:1-7.
Shah et al. (1983) J. Biol. Chem. 258:12064-12068.
Shah et al. (1999). J. Bacteriol. 181:2797-2801.
Siddavattam et al. (1993). Mol. Gen. Genet. 239:435-440.
Sippel and Einsle, (2017). Nature Chemical Biology, 13:956. doi:10.1038/nchembio.2428
Sippel et al. (2018). Journal of Biological Inorganic Chemistry 23(7): 1049-1056
Sirrenberg et al. (1996). Nature 384: 6609 582-5.
Smil (2002). Ambio 31:126-131.
Smith et al. (1997). J. Bacteriol. 179:7135-7155.
Smith et al. (2005). Annu. Rev. Biochem. 74:247-281.
Smanski et al. (2014). Nature Biotechnology 32:1241-1249.
Spatzal et al. (2016). Nature communications 7 (2016): 10902,
Stalker et al. (1988). J. Biol. Chem. 263(13):6310-6314.
Staples et al. (2007). J. Bacteriol. 189:7392-7398.
Suh et al. (2003). Journal of Biological Chemistry 278:5353-5360.
Sutton et al. (2008). Environ Pollut 156:583-604.
Temme et al. (2012). Proc. Natl. Acad. Sci. U.S.A. 109(18):7085-7090.
Tezcan et al. (2005). Science 309:1377-1380.
Thiel et al. (1995) Proc Natl Acad Sci U S A 92: 9358-9362.
Thiel et al. (1997). J. Bacteriol. 179:5222-5225.
Thillet et al. (1988). J. Biol. Chem 263(25):12500-12508
Thomas et al. (1966). Biochemistry 5(8):2513-2516.
Toriyama et al. (1986). Theor. Appl. Genet. 73:16-19
Verhasselt et al. (1995). Yeast 11(10):961-966.
von Heijne (1986). EMBO J. 5:1335-1342.
Wagner et al. (1992). Proc. Nail. Acad. Sci. U.S.A. 89:6099-6103
Wahlund и Madigan, (1993). J. Bacteriol. 175: 474-478.
Wang et al. (2013). PLoS Genet 9, e1003865.
Waterhouse et al. (2018). Nucleic Acids Res. 46(W1), W296-W303.
Weber et al. (2011) PloS one. 6(2), pp.e16765.
Wiig et al. (2011) Proc Natl Acad Sci USA 108: 8623-8627.
Wisniewski et al. (2011) Anal Biochem. 410:307-9.
Wood et al. (2009). Plant Biotechnol J. 7:914-924.
Xia et al. (2009). Nucleic acids research 37(suppl_2): W652-W660.
Xiao et al. (2010). Biochemistry 49:5588-5599.
Yang et al. (2014). Proc. Natl. Acad. Sci. U.S.A. 111:E3718-E3725.
Yang et al. (2010). BMC Plant Biology, 10. doi:10.1186/1471-2229-10-231.
Yang et al. (2017). Proc Natl Acad Sci USA 114:E2460-E2465.
Yang et al. (2018) Proc Natl Acad Sci USA doi/10.1073/pnas.1804992115
Yates (1972) FEBS Lett 27:63-67.
Yoneda et al. (2012). Int. J. Systematic Evol. Biol. 62:1692-1697.
Yuvaniyama et al. (2000) Proc. Natl. Acad. Sci. USA 97:599-604.
Zhang and Glaser (2002). Trends Plant Sci 7:14-21.
Zhang et al. (2009). Progress in Natural Science 19:1197-1200.
Zhang et al. (2016). Biogeochemistry 127, 189-198. doi: 10.1007/s10533-016-0188-6.
Zhang and Wang (2013). PLoS One 8(7). doi:10.1371/journal.pone.0068491.
Zheng et al. (1994)
Zheng et al. (1997). J. Bacteriol. 179:5963-5966.
Zheng et al. (2018). Nature Microbiology, 3:281-286. doi:10.1038/s41564-017-0091-5.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Государственное объединение научных и прикладных исследований
<120> ЭКСПРЕССИЯ ПОЛИПЕПТИДОВ НИТРОГЕНАЗЫ В РАСТИТЕЛЬНЫХ КЛЕТКАХ
<130> 529584PCT
<150> AU 2020900689
<151> 2020-03-05
<150> AU 2019903818
<151> 2019-10-10
<150> AU 2019900780
<151> 2019-03-08
<160> 248
<170> Версия PatentIn 3.5
<210> 1
<211> 293
<212> PRT
<213> Klebsiella oxytoca
<400> 1
Met Thr Met Arg Gln Cys Ala Ile Tyr Gly Lys Gly Gly Ile Gly Lys
1. 5 10 15
Ser Thr Thr Thr Gln Asn Leu Val Ala Ala Leu Ala Glu Met Gly Lys
20 25 30
Lys Val Met Ile Val Gly Cys Asp Pro Lys Ala Asp Ser Thr Arg Leu
35 40 45
Ile Leu His Ala Lys Ala Gln Asn Thr Ile Met Glu Met Ala Ala Glu
50 55 60
Val Gly Ser Val Glu Asp Leu Glu Leu Glu Asp Val Leu Gln Ile Gly
65 70 75 80
Tyr Gly Asp Val Arg Cys Ala Glu Ser Gly Gly Pro Glu Pro Gly Val
85 90 95
Gly Cys Ala Gly Arg Gly Val Ile Thr Ala Ile Asn Phe Leu Glu Glu
100 105 110
Glu Gly Ala Tyr Glu Asp Asp Leu Asp Phe Val Phe Tyr Asp Val Leu
115 120 125
Gly Asp Val Val Cys Gly Gly Phe Ala Met Pro Ile Arg Glu Asn Lys
130 135 140
Ala Gln Glu Ile Tyr Ile Val Cys Ser Gly Glu Met Met Ala Met Tyr
145 150 155 160
Ala Ala Asn Asn Ile Ser Lys Gly Ile Val Lys Tyr Ala Lys Ser Gly
165 170 175
Lys Val Arg Leu Gly Gly Leu Ile Cys Asn Ser Arg Gln Thr Asp Arg
180 185 190
Glu Asp Glu Leu Ile Ile Ala Leu Ala Glu Lys Leu Gly Thr Gln Met
195 200 205
Ile His Phe Val Pro Arg Asp Asn Ile Val Gln Arg Ala Glu Ile Arg
210 215 220
Arg Met Thr Val Ile Glu Tyr Asp Pro Ala Cys Lys Gln Ala Asn Glu
225 230 235 240
Tyr Arg Thr Leu Ala Gln Lys Ile Val Asn Asn Thr Met Lys Val Val
245 250 255
Pro Thr Pro Cys Thr Met Asp Glu Leu Glu Ser Leu Leu Met Glu Phe
260 265 270
Gly Ile Met Glu Glu Glu Asp Thr Ser Ile Ile Gly Lys Thr Ala Ala
275 280 285
Glu Glu Asn Ala Ala
290
<210> 2
<211> 483
<212> PRT
<213> Klebsiella oxytoca
<400> 2
Met Met Thr Asn Ala Thr Gly Glu Arg Asn Leu Ala Leu Ile Gln Glu
1. 5 10 15
Val Leu Glu Val Phe Pro Glu Thr Ala Arg Lys Glu Arg Arg Lys His
20 25 30
Met Met Val Ser Asp Pro Lys Met Lys Ser Val Gly Lys Cys Ile Ile
35 40 45
Ser Asn Arg Lys Ser Gln Pro Gly Val Met Thr Val Arg Gly Cys Ala
50 55 60
Tyr Ala Gly Ser Lys Gly Val Val Phe Gly Pro Ile Lys Asp Met Ala
65 70 75 80
His Ile Ser His Gly Pro Ala Gly Cys Gly Gln Tyr Ser Arg Ala Glu
85 90 95
Arg Arg Asn Tyr Tyr Thr Gly Val Ser Gly Val Asp Ser Phe Gly Thr
100 105 110
Leu Asn Phe Thr Ser Asp Phe Gln Glu Arg Asp Ile Val Phe Gly Gly
115 120 125
Asp Lys Lys Leu Ser Lys Leu Ile Glu Glu Met Glu Leu Leu Phe Pro
130 135 140
Leu Thr Lys Gly Ile Thr Ile Gln Ser Glu Cys Pro Val Gly Leu Ile
145 150 155 160
Gly Asp Asp Ile Ser Ala Val Ala Asn Ala Ser Ser Lys Ala Leu Asp
165 170 175
Lys Pro Val Ile Pro Val Arg Cys Glu Gly Phe Arg Gly Val Ser Gln
180 185 190
Ser Leu Gly His His Ile Ala Asn Asp Val Val Arg Asp Trp Ile Leu
195 200 205
Asn Asn Arg Glu Gly Gln Pro Phe Glu Thr Thr Pro Tyr Asp Val Ala
210 215 220
Ile Ile Gly Asp Tyr Asn Ile Gly Gly Asp Ala Trp Ala Ser Arg Ile
225 230 235 240
Leu Leu Glu Glu Met Gly Leu Arg Val Val Ala Gln Trp Ser Gly Asp
245 250 255
Gly Thr Leu Val Glu Met Glu Asn Thr Pro Phe Val Lys Leu Asn Leu
260 265 270
Val His Cys Tyr Arg Ser Met Asn Tyr Ile Ala Arg His Met Glu Glu
275 280 285
Lys His Gln Ile Pro Trp Met Glu Tyr Asn Phe Phe Gly Pro Thr Lys
290 295 300
Ile Ala Glu Ser Leu Arg Lys Ile Ala Asp Gln Phe Asp Asp Thr Ile
305 310 315 320
Arg Ala Asn Ala Glu Ala Val Ile Ala Arg Tyr Glu Gly Gln Met Ala
325 330 335
Ala Ile Ile Ala Lys Tyr Arg Pro Arg Leu Glu Gly Arg Lys Val Leu
340 345 350
Leu Tyr Ile Gly Gly Leu Arg Pro Arg His Val Ile Gly Ala Tyr Glu
355 360 365
Asp Leu Gly Met Glu Ile Ile Ala Ala Gly Tyr Glu Phe Ala His Asn
370 375 380
Asp Asp Tyr Asp Arg Thr Leu Pro Asp Leu Lys Glu Gly Thr Leu Leu
385 390 395 400
Phe Asp Asp Ala Ser Ser Tyr Glu Leu Glu Ala Phe Val Lys Ala Leu
405 410 415
Lys Pro Asp Leu Ile Gly Ser Gly Ile Lys Glu Lys Tyr Ile Phe Gln
420 425 430
Lys Met Gly Val Pro Phe Arg Gln Met His Ser Trp Asp Tyr Ser Gly
435 440 445
Pro Tyr His Gly Tyr Asp Gly Phe Ala Ile Phe Ala Arg Asp Met Asp
450 455 460
Met Thr Leu Asn Asn Pro Ala Trp Asn Glu Leu Thr Ala Pro Trp Leu
465 470 475 480
Lys Ser Ala
<210> 3
<211> 520
<212> PRT
<213> Klebsiella oxytoca
<400> 3
Met Ser Gln Thr Ile Asp Lys Ile Asn Ser Cys Tyr Pro Leu Phe Glu
1. 5 10 15
Gln Asp Glu Tyr Gln Glu Leu Phe Arg Asn Lys Arg Gln Leu Glu Glu
20 25 30
Ala His Asp Ala Gln Arg Val Gln Glu Val Phe Ala Trp Thr Thr Thr
35 40 45
Ala Glu Tyr Glu Ala Leu Asn Phe Gln Arg Glu Ala Leu Thr Val Asp
50 55 60
Pro Ala Lys Ala Cys Gln Pro Leu Gly Ala Val Leu Cys Ser Leu Gly
65 70 75 80
Phe Ala Asn Thr Leu Pro Tyr Val His Gly Ser Gln Gly Cys Val Ala
85 90 95
Tyr Phe Arg Thr Tyr Phe Asn Arg His Phe Lys Glu Pro Ile Ala Cys
100 105 110
Val Ser Asp Ser Met Thr Glu Asp Ala Ala Val Phe Gly Gly Asn Asn
115 120 125
Asn Met Asn Leu Gly Leu Gln Asn Ala Ser Ala Leu Tyr Lys Pro Glu
130 135 140
Ile Ile Ala Val Ser Thr Thr Cys Met Ala Glu Val Ile Gly Asp Asp
145 150 155 160
Leu Gln Ala Phe Ile Ala Asn Ala Lys Lys Asp Gly Phe Val Asp Ser
165 170 175
Ser Ile Ala Val Pro His Ala His Thr Pro Ser Phe Ile Gly Ser His
180 185 190
Val Thr Gly Trp Asp Asn Met Phe Glu Gly Phe Ala Lys Thr Phe Thr
195 200 205
Ala Asp Tyr Gln Gly Gln Pro Gly Lys Leu Pro Lys Leu Asn Leu Val
210 215 220
Thr Gly Phe Glu Thr Tyr Leu Gly Asn Phe Arg Val Leu Lys Arg Met
225 230 235 240
Met Glu Gln Met Ala Val Pro Cys Ser Leu Leu Ser Asp Pro Ser Glu
245 250 255
Val Leu Asp Thr Pro Ala Asp Gly His Tyr Arg Met Tyr Ser Gly Gly
260 265 270
Thr Thr Gln Gln Glu Met Lys Glu Ala Pro Asp Ala Ile Asp Thr Leu
275 280 285
Leu Leu Gln Pro Trp Gln Leu Leu Lys Ser Lys Lys Val Val Gln Glu
290 295 300
Met Trp Asn Gln Pro Ala Thr Glu Val Ala Ile Pro Leu Gly Leu Ala
305 310 315 320
Ala Thr Asp Glu Leu Leu Met Thr Val Ser Gln Leu Ser Gly Lys Pro
325 330 335
Ile Ala Asp Ala Leu Thr Leu Glu Arg Gly Arg Leu Val Asp Met Met
340 345 350
Leu Asp Ser His Thr Trp Leu His Gly Lys Lys Phe Gly Leu Tyr Gly
355 360 365
Asp Pro Asp Phe Val Met Gly Leu Thr Arg Phe Leu Leu Glu Leu Gly
370 375 380
Cys Glu Pro Thr Val Ile Leu Ser His Asn Ala Asn Lys Arg Trp Gln
385 390 395 400
Lys Ala Met Asn Lys Met Leu Asp Ala Ser Pro Tyr Gly Arg Asp Ser
405 410 415
Glu Val Phe Ile Asn Cys Asp Leu Trp His Phe Arg Ser Leu Met Phe
420 425 430
Thr Arg Gln Pro Asp Phe Met Ile Gly Asn Ser Tyr Gly Lys Phe Ile
435 440 445
Gln Arg Asp Thr Leu Ala Lys Gly Lys Ala Phe Glu Val Pro Leu Ile
450 455 460
Arg Leu Gly Phe Pro Leu Phe Asp Arg His His Leu His Arg Gln Thr
465 470 475 480
Thr Trp Gly Tyr Glu Gly Ala Met Asn Ile Val Thr Thr Leu Val Asn
485 490 495
Ala Val Leu Glu Lys Leu Asp Ser Asp Thr Ser Gln Leu Gly Lys Thr
500 505 510
Asp Tyr Ser Phe Asp Leu Val Arg
515 520
<210> 4
<211> 468
<212> PRT
<213> Klebsiella oxytoca
<400> 4
Met Thr Ser Cys Ser Ser Phe Ser Gly Gly Lys Ala Cys Arg Pro Ala
1. 5 10 15
Asp Asp Ser Ala Leu Thr Pro Leu Val Ala Asp Lys Ala Ala Ala His
20 25 30
Pro Cys Tyr Ser Arg His Gly His His Arg Phe Ala Arg Met His Leu
35 40 45
Pro Val Ala Pro Ala Cys Asn Leu Gln Cys Asn Tyr Cys Asn Arg Lys
50 55 60
Phe Asp Cys Ser Asn Glu Ser Arg Pro Gly Val Ser Ser Thr Leu Leu
65 70 75 80
Thr Pro Glu Gln Ala Val Val Lys Val Arg Gln Val Ala Gln Ala Ile
85 90 95
Pro Gln Leu Ser Val Val Gly Ile Ala Gly Pro Gly Asp Pro Leu Ala
100 105 110
Asn Ile Ala Arg Thr Phe Arg Thr Leu Glu Leu Ile Arg Glu Gln Leu
115 120 125
Pro Asp Leu Lys Leu Cys Leu Ser Thr Asn Gly Leu Val Leu Pro Asp
130 135 140
Ala Val Asp Arg Leu Leu Asp Val Gly Val Asp His Val Thr Val Thr
145 150 155 160
Ile Asn Thr Leu Asp Ala Glu Ile Ala Ala Gln Ile Tyr Ala Trp Leu
165 170 175
Trp Leu Asp Gly Glu Arg Tyr Ser Gly Arg Glu Ala Gly Glu Ile Leu
180 185 190
Ile Ala Arg Gln Leu Glu Gly Val Arg Arg Leu Thr Ala Lys Gly Val
195 200 205
Leu Val Lys Ile Asn Ser Val Leu Ile Pro Gly Ile Asn Asp Ser Gly
210 215 220
Met Ala Gly Val Ser Arg Ala Leu Arg Ala Ser Gly Ala Phe Ile His
225 230 235 240
Asn Ile Met Pro Leu Ile Ala Arg Pro Glu His Gly Thr Val Phe Gly
245 250 255
Leu Asn Gly Gln Pro Glu Pro Asp Ala Glu Thr Leu Ala Ala Thr Arg
260 265 270
Ser Arg Cys Gly Glu Val Met Pro Gln Met Thr His Cys His Gln Cys
275 280 285
Arg Ala Asp Ala Ile Gly Met Leu Gly Glu Asp Arg Ser Gln Gln Phe
290 295 300
Thr Gln Leu Pro Ala Pro Glu Ser Leu Pro Ala Trp Leu Pro Ile Leu
305 310 315 320
His Gln Arg Ala Gln Leu His Ala Ser Ile Ala Thr Arg Gly Glu Ser
325 330 335
Glu Ala Asp Asp Ala Cys Leu Val Ala Val Ala Ser Ser Arg Gly Asp
340 345 350
Val Ile Asp Cys His Phe Gly His Ala Asp Arg Phe Tyr Ile Tyr Ser
355 360 365
Leu Ser Ala Ala Gly Met Val Leu Val Asn Glu Arg Phe Thr Pro Lys
370 375 380
Tyr Cys Gln Gly Arg Asp Asp Cys Glu Pro Gln Asp Asn Ala Ala Arg
385 390 395 400
Phe Ala Ala Ile Leu Glu Leu Leu Ala Asp Val Lys Ala Val Phe Cys
405 410 415
Val Arg Ile Gly His Thr Pro Trp Gln Gln Leu Glu Gln Glu Gly Ile
420 425 430
Glu Pro Cys Val Asp Gly Ala Trp Arg Pro Val Ser Glu Val Leu Pro
435 440 445
Ala Trp Trp Gln Gln Arg Arg Gly Ser Trp Pro Ala Ala Leu Pro His
450 455 460
Lys Gly Val Ala
465
<210> 5
<211> 457
<212> PRT
<213> Klebsiella oxytoca
<400> 5
Met Lys Gly Asn Glu Ile Leu Ala Leu Leu Asp Glu Pro Ala Cys Glu
1. 5 10 15
His Asn His Lys Gln Lys Ser Gly Cys Ser Ala Pro Lys Pro Gly Ala
20 25 30
Thr Ala Ala Gly Cys Ala Phe Asp Gly Ala Gln Ile Thr Leu Leu Pro
35 40 45
Ile Ala Asp Val Ala His Leu Val His Gly Pro Ile Gly Cys Ala Gly
50 55 60
Ser Ser Trp Asp Asn Arg Gly Ser Ala Ser Ser Gly Pro Thr Leu Asn
65 70 75 80
Arg Leu Gly Phe Thr Thr Asp Leu Asn Glu Gln Asp Val Ile Met Gly
85 90 95
Arg Gly Glu Arg Arg Leu Phe His Ala Val Arg His Ile Val Thr Arg
100 105 110
Tyr His Pro Ala Ala Val Phe Ile Tyr Asn Thr Cys Val Pro Ala Met
115 120 125
Glu Gly Asp Asp Leu Glu Ala Val Cys Gln Ala Ala Gln Thr Ala Thr
130 135 140
Gly Val Pro Val Ile Ala Ile Asp Ala Ala Gly Phe Tyr Gly Ser Lys
145 150 155 160
Asn Leu Gly Asn Arg Pro Ala Gly Asp Val Met Val Lys Arg Val Ile
165 170 175
Gly Gln Arg Glu Pro Ala Pro Trp Pro Glu Ser Thr Leu Phe Ala Pro
180 185 190
Glu Gln Arg His Asp Ile Gly Leu Ile Gly Glu Phe Asn Ile Ala Gly
195 200 205
Glu Phe Trp His Ile Gln Pro Leu Leu Asp Glu Leu Gly Ile Arg Val
210 215 220
Leu Gly Ser Leu Ser Gly Asp Gly Arg Phe Ala Glu Ile Gln Thr Met
225 230 235 240
His Arg Ala Gln Ala Asn Met Leu Val Cys Ser Arg Ala Leu Ile Asn
245 250 255
Val Ala Arg Ala Leu Glu Gln Arg Tyr Gly Thr Pro Trp Phe Glu Gly
260 265 270
Ser Phe Tyr Gly Ile Arg Ala Thr Ser Asp Ala Leu Arg Gln Leu Ala
275 280 285
Ala Leu Leu Gly Asp Asp Asp Leu Arg Gln Arg Thr Glu Ala Leu Ile
290 295 300
Ala Arg Glu Glu Gln Ala Ala Glu Leu Ala Leu Gln Pro Trp Arg Glu
305 310 315 320
Gln Leu Arg Gly Arg Lys Ala Leu Leu Tyr Thr Gly Gly Val Lys Ser
325 330 335
Trp Ser Val Val Ser Ala Leu Gln Asp Leu Gly Met Thr Val Val Ala
340 345 350
Thr Gly Thr Arg Lys Ser Thr Glu Glu Asp Lys Gln Arg Ile Arg Glu
355 360 365
Leu Met Gly Glu Glu Ala Val Met Leu Glu Glu Gly Asn Ala Arg Thr
370 375 380
Leu Leu Asp Val Val Tyr Arg Tyr Gln Ala Asp Leu Met Ile Ala Gly
385 390 395 400
Gly Arg Asn Met Tyr Thr Ala Tyr Lys Ala Arg Leu Pro Phe Leu Asp
405 410 415
Ile Asn Gln Glu Arg Glu His Ala Phe Ala Gly Tyr Gln Gly Ile Val
420 425 430
Thr Leu Ala Arg Gln Leu Cys Gln Thr Ile Asn Ser Pro Ile Trp Pro
435 440 445
Gln Thr His Ser Arg Ala Pro Trp Arg
450 455
<210> 6
<211> 176
<212> PRT
<213> Klebsiella oxytoca
<400> 6
Met Ala Asn Ile Gly Ile Phe Phe Gly Thr Asp Thr Gly Lys Thr Arg
1. 5 10 15
Lys Ile Ala Lys Met Ile His Lys Gln Leu Gly Glu Leu Ala Asp Ala
20 25 30
Pro Val Asn Ile Asn Arg Thr Thr Leu Asp Asp Phe Met Ala Tyr Pro
35 40 45
Val Leu Leu Leu Gly Thr Pro Thr Leu Gly Asp Gly Gln Leu Pro Gly
50 55 60
Leu Glu Ala Gly Cys Glu Ser Glu Ser Trp Ser Glu Phe Ile Ser Gly
65 70 75 80
Leu Asp Asp Ala Ser Leu Lys Gly Lys Thr Val Ala Leu Phe Gly Leu
85 90 95
Gly Asp Gln Arg Gly Tyr Pro Asp Asn Phe Val Ser Gly Met Arg Pro
100 105 110
Leu Phe Asp Ala Leu Ser Ala Arg Gly Ala Gln Met Ile Gly Ser Trp
115 120 125
Pro Asn Glu Gly Tyr Glu Phe Ser Ala Ser Ser Ala Leu Glu Gly Asp
130 135 140
Arg Phe Val Gly Leu Val Leu Asp Gln Asp Asn Gln Phe Asp Gln Thr
145 150 155 160
Glu Ala Arg Leu Ala Ser Trp Leu Glu Glu Ile Lys Arg Thr Val Leu
165 170 175
<210> 7
<211> 1171
<212> PRT
<213> Klebsiella oxytoca
<400> 7
Met Ser Gly Lys Met Lys Thr Met Asp Gly Asn Ala Ala Ala Ala Trp
1. 5 10 15
Ile Ser Tyr Ala Phe Thr Glu Val Ala Ala Ile Tyr Pro Ile Thr Pro
20 25 30
Ser Thr Pro Met Ala Glu Asn Val Asp Glu Trp Ala Ala Gln Gly Lys
35 40 45
Lys Asn Leu Phe Gly Gln Pro Val Arg Leu Met Glu Met Gln Ser Glu
50 55 60
Ala Gly Ala Ala Gly Ala Val His Gly Ala Leu Gln Ala Gly Ala Leu
65 70 75 80
Thr Thr Thr Tyr Thr Ala Ser Gln Gly Leu Leu Leu Met Ile Pro Asn
85 90 95
Met Tyr Lys Ile Ala Gly Glu Leu Leu Pro Gly Val Phe His Val Ser
100 105 110
Ala Arg Ala Leu Ala Thr Asn Ser Leu Asn Ile Phe Gly Asp His Gln
115 120 125
Asp Val Met Ala Val Arg Gln Thr Gly Cys Ala Met Leu Ala Glu Asn
130 135 140
Asn Val Gln Gln Val Met Asp Leu Ser Ala Val Ala His Leu Ala Ala
145 150 155 160
Ile Lys Gly Arg Ile Pro Phe Val Asn Phe Phe Asp Gly Phe Arg Thr
165 170 175
Ser His Glu Ile Gln Lys Ile Glu Val Leu Glu Tyr Glu Gln Leu Ala
180 185 190
Thr Leu Leu Asp Arg Pro Ala Leu Asp Ser Phe Arg Arg Asn Ala Leu
195 200 205
His Pro Asp His Pro Val Ile Arg Gly Thr Ala Gln Asn Pro Asp Ile
210 215 220
Tyr Phe Gln Glu Arg Glu Ala Gly Asn Arg Phe Tyr Gln Ala Leu Pro
225 230 235 240
Asp Ile Val Glu Ser Tyr Met Thr Gln Ile Ser Ala Leu Thr Gly Arg
245 250 255
Glu Tyr His Leu Phe Asn Tyr Thr Gly Ala Ala Asp Ala Glu Arg Val
260 265 270
Ile Ile Ala Met Gly Ser Val Cys Asp Thr Val Gln Glu Val Val Asp
275 280 285
Thr Leu Asn Ala Ala Gly Glu Lys Val Gly Leu Leu Ser Val His Leu
290 295 300
Phe Arg Pro Phe Ser Leu Ala His Phe Phe Ala Gln Leu Pro Lys Thr
305 310 315 320
Val Gln Arg Ile Ala Val Leu Asp Arg Thr Lys Glu Pro Gly Ala Gln
325 330 335
Ala Glu Pro Leu Cys Leu Asp Val Lys Asn Ala Phe Tyr His His Asp
340 345 350
Asp Ala Pro Leu Ile Val Gly Gly Arg Tyr Ala Leu Gly Gly Lys Asp
355 360 365
Val Leu Pro Asn Asp Ile Ala Ala Val Phe Asp Asn Leu Asn Lys Pro
370 375 380
Leu Pro Met Asp Gly Phe Thr Leu Gly Ile Val Asp Asp Val Thr Phe
385 390 395 400
Thr Ser Leu Pro Pro Arg Gln Gln Thr Leu Ala Val Ser His Asp Gly
405 410 415
Ile Thr Ala Cys Lys Phe Trp Gly Met Gly Ser Asp Gly Thr Val Gly
420 425 430
Ala Asn Lys Ser Ala Ile Lys Ile Ile Gly Asp Lys Thr Pro Leu Tyr
435 440 445
Ala Gln Ala Tyr Phe Ser Tyr Asp Ser Lys Lys Ser Gly Gly Ile Thr
450 455 460
Val Ser His Leu Arg Phe Gly Asp Arg Pro Ile Asn Ser Pro Tyr Leu
465 470 475 480
Ile His Arg Ala Asp Phe Ile Ser Cys Ser Gln Gln Ser Tyr Val Glu
485 490 495
Arg Tyr Asp Leu Leu Asp Gly Leu Lys Pro Gly Gly Thr Phe Leu Leu
500 505 510
Asn Cys Ser Trp Ser Asp Ala Glu Leu Glu Gln His Leu Pro Val Gly
515 520 525
Phe Lys Arg Tyr Leu Ala Arg Glu Asn Ile His Phe Tyr Thr Leu Asn
530 535 540
Ala Val Asp Ile Ala Arg Glu Leu Gly Leu Gly Gly Arg Phe Asn Met
545 550 555 560
Leu Met Gln Ala Ala Phe Phe Lys Leu Ala Ala Ile Ile Asp Pro Gln
565 570 575
Thr Ala Ala Asp Tyr Leu Lys Gln Ala Val Glu Lys Ser Tyr Gly Ser
580 585 590
Lys Gly Ala Ala Val Ile Glu Met Asn Gln Arg Ala Ile Glu Leu Gly
595 600 605
Met Ala Ser Leu His Gln Val Thr Ile Pro Ala His Trp Ala Thr Leu
610 615 620
Asp Glu Pro Ala Ala Gln Ala Ser Ala Met Met Pro Asp Phe Ile Arg
625 630 635 640
Asp Ile Leu Gln Pro Met Asn Arg Gln Cys Gly Asp Gln Leu Pro Val
645 650 655
Ser Ala Phe Val Gly Met Glu Asp Gly Thr Phe Pro Ser Gly Thr Ala
660 665 670
Ala Trp Glu Lys Arg Gly Ile Ala Leu Glu Val Pro Val Trp Gln Pro
675 680 685
Glu Gly Cys Thr Gln Cys Asn Gln Cys Ala Phe Ile Cys Pro His Ala
690 695 700
Ala Ile Arg Pro Ala Leu Leu Asn Gly Glu Glu His Asp Ala Ala Pro
705 710 715 720
Val Gly Leu Leu Ser Lys Pro Ala Gln Gly Ala Lys Glu Tyr His Tyr
725 730 735
His Leu Ala Ile Ser Pro Leu Asp Cys Ser Gly Cys Gly Asn Cys Val
740 745 750
Asp Ile Cys Pro Ala Arg Gly Lys Ala Leu Lys Met Gln Ser Leu Asp
755 760 765
Ser Gln Arg Gln Met Ala Pro Val Trp Asp Tyr Ala Leu Ala Leu Thr
770 775 780
Pro Lys Ser Asn Pro Phe Arg Lys Thr Thr Val Lys Gly Ser Gln Phe
785 790 795 800
Glu Thr Pro Leu Leu Glu Phe Ser Gly Ala Cys Ala Gly Cys Gly Glu
805 810 815
Thr Pro Tyr Ala Arg Leu Ile Thr Gln Leu Phe Gly Asp Arg Met Leu
820 825 830
Ile Ala Asn Ala Thr Gly Cys Ser Ser Ile Trp Gly Ala Ser Ala Pro
835 840 845
Ser Ile Pro Tyr Thr Thr Asn His Arg Gly His Gly Pro Ala Trp Ala
850 855 860
Asn Ser Leu Phe Glu Asp Asn Ala Glu Phe Gly Leu Gly Met Met Leu
865 870 875 880
Gly Gly Gln Ala Val Arg Gln Gln Ile Ala Asp Asp Met Thr Ala Ala
885 890 895
Leu Ala Leu Pro Val Ser Asp Glu Leu Ser Asp Ala Met Arg Gln Trp
900 905 910
Leu Ala Lys Gln Asp Glu Gly Glu Gly Thr Arg Glu Arg Ala Asp Arg
915 920 925
Leu Ser Glu Arg Leu Ala Ala Glu Lys Glu Gly Val Pro Leu Leu Glu
930 935 940
Gln Leu Trp Gln Asn Arg Asp Tyr Phe Val Arg Arg Ser Gln Trp Ile
945 950 955 960
Phe Gly Gly Asp Gly Trp Ala Tyr Asp Ile Gly Phe Gly Gly Leu Asp
965 970 975
His Val Leu Ala Ser Gly Glu Asp Val Asn Ile Leu Val Phe Asp Thr
980 985 990
Glu Val Tyr Ser Asn Thr Gly Gly Gln Ser Ser Lys Ser Thr Pro Val
995 1000 1005
Ala Ala Ile Ala Lys Phe Ala Ala Gln Gly Lys Arg Thr Arg Lys
1010 1015 1020
Lys Asp Leu Gly Met Met Ala Met Ser Tyr Gly Asn Val Tyr Val
1025 1030 1035
Ala Gln Val Ala Met Gly Ala Asp Lys Asp Gln Thr Leu Arg Ala
1040 1045 1050
Ile Ala Glu Ala Glu Ala Trp Pro Gly Pro Ser Leu Val Ile Ala
1055 1060 1065
Tyr Ala Ala Cys Ile Asn His Gly Leu Lys Ala Gly Met Arg Cys
1070 1075 1080
Ser Gln Arg Glu Ala Lys Arg Ala Val Glu Ala Gly Tyr Trp His
1085 1090 1095
Leu Trp Arg Tyr His Pro Gln Arg Glu Ala Glu Gly Lys Thr Pro
1100 1105 1110
Phe Met Leu Asp Ser Glu Glu Pro Glu Glu Ser Phe Arg Asp Phe
1115 1120 1125
Leu Leu Gly Glu Val Arg Tyr Ala Ser Leu His Lys Thr Thr Pro
1130 1135 1140
His Leu Ala Asp Ala Leu Phe Ser Arg Thr Glu Glu Asp Ala Arg
1145 1150 1155
Ala Arg Phe Ala Gln Tyr Arg Arg Leu Ala Gly Glu Glu
1160 1165 1170
<210> 8
<211> 266
<212> PRT
<213> Klebsiella oxytoca
<400> 8
Met Asn Pro Trp Gln Arg Phe Ala Arg Gln Arg Leu Ala Arg Ser Arg
1. 5 10 15
Trp Asn Arg Asp Pro Ala Ala Leu Asp Pro Ala Asp Thr Pro Ala Phe
20 25 30
Glu Gln Ala Trp Gln Arg Gln Cys His Met Glu Gln Thr Ile Val Ala
35 40 45
Arg Val Pro Glu Gly Asp Ile Pro Ala Ala Leu Leu Glu Asn Ile Ala
50 55 60
Ala Ser Leu Ala Ile Trp Leu Asp Glu Gly Asp Phe Ala Pro Pro Glu
65 70 75 80
Arg Ala Ala Ile Val Arg His His Ala Arg Leu Glu Leu Ala Phe Ala
85 90 95
Asp Ile Ala Arg Gln Ala Pro Gln Pro Asp Leu Ser Thr Val Gln Ala
100 105 110
Trp Tyr Leu Arg His Gln Thr Gln Phe Met Arg Pro Glu Gln Arg Leu
115 120 125
Thr Arg His Leu Leu Leu Thr Val Asp Asn Asp Arg Glu Ala Val His
130 135 140
Gln Arg Ile Leu Gly Leu Tyr Arg Gln Ile Asn Ala Ser Arg Asp Ala
145 150 155 160
Phe Ala Pro Leu Ala Gln Arg His Ser His Cys Pro Ser Ala Leu Glu
165 170 175
Glu Gly Arg Leu Gly Trp Ile Ser Arg Gly Leu Leu Tyr Pro Gln Leu
180 185 190
Glu Thr Ala Leu Phe Ser Leu Ala Glu Asn Ala Leu Ser Leu Pro Ile
195 200 205
Ala Ser Glu Leu Gly Trp His Leu Leu Trp Cys Glu Ala Ile Arg Pro
210 215 220
Ala Ala Pro Met Glu Pro Gln Gln Ala Leu Glu Ser Ala Arg Asp Tyr
225 230 235 240
Leu Trp Gln Gln Ser Gln Gln Arg His Gln Arg Gln Trp Leu Glu Gln
245 250 255
Met Ile Ser Arg Gln Pro Gly Leu Cys Gly
260 265
<210> 9
<211> 461
<212> PRT
<213> Klebsiella oxytoca
<400> 9
Met Ala Asp Ile Phe Arg Thr Asp Lys Pro Leu Ala Val Ser Pro Ile
1. 5 10 15
Lys Thr Gly Gln Pro Leu Gly Ala Ile Leu Ala Ser Leu Gly Ile Glu
20 25 30
His Ser Ile Pro Leu Val His Gly Ala Gln Gly Cys Ser Ala Phe Ala
35 40 45
Lys Val Phe Phe Ile Gln His Phe His Asp Pro Val Pro Leu Gln Ser
50 55 60
Thr Ala Met Asp Pro Thr Ser Thr Ile Met Gly Ala Asp Gly Asn Ile
65 70 75 80
Phe Thr Ala Leu Asp Thr Leu Cys Gln Arg Asn Asn Pro Gln Ala Ile
85 90 95
Val Leu Leu Ser Thr Gly Leu Ser Glu Ala Gln Gly Ser Asp Ile Ser
100 105 110
Arg Val Val Arg Gln Phe Arg Glu Glu Tyr Pro Arg His Lys Gly Val
115 120 125
Ala Ile Leu Thr Val Asn Thr Pro Asp Phe Tyr Gly Ser Met Glu Asn
130 135 140
Gly Phe Ser Ala Val Leu Glu Ser Val Ile Glu Gln Trp Val Pro Pro
145 150 155 160
Ala Pro Arg Pro Ala Gln Arg Asn Arg Arg Val Asn Leu Leu Val Ser
165 170 175
His Leu Cys Ser Pro Gly Asp Ile Glu Trp Leu Arg Arg Cys Val Glu
180 185 190
Ala Phe Gly Leu Gln Pro Ile Ile Leu Pro Asp Leu Ala Gln Ser Met
195 200 205
Asp Gly His Leu Ala Gln Gly Asp Phe Ser Pro Leu Thr Gln Gly Gly
210 215 220
Thr Pro Leu Arg Gln Ile Glu Gln Met Gly Gln Ser Leu Cys Ser Phe
225 230 235 240
Ala Ile Gly Val Ser Leu His Arg Ala Ser Ser Leu Leu Ala Pro Arg
245 250 255
Cys Arg Gly Glu Val Ile Ala Leu Pro His Leu Met Thr Leu Glu Arg
260 265 270
Cys Asp Ala Phe Ile His Gln Leu Ala Lys Ile Ser Gly Arg Ala Val
275 280 285
Pro Glu Trp Leu Glu Arg Gln Arg Gly Gln Leu Gln Asp Ala Met Ile
290 295 300
Asp Cys His Met Trp Leu Gln Gly Gln Arg Met Ala Ile Ala Ala Glu
305 310 315 320
Gly Asp Leu Leu Ala Ala Trp Cys Asp Phe Ala Asn Ser Gln Gly Met
325 330 335
Gln Pro Gly Pro Leu Val Ala Pro Thr Gly His Pro Ser Leu Arg Gln
340 345 350
Leu Pro Val Glu Arg Val Val Pro Gly Asp Leu Glu Asp Leu Gln Thr
355 360 365
Leu Leu Cys Ala His Pro Ala Asp Leu Leu Val Ala Asn Ser His Ala
370 375 380
Arg Asp Leu Ala Glu Gln Phe Ala Leu Pro Leu Val Arg Ala Gly Phe
385 390 395 400
Pro Leu Phe Asp Lys Leu Gly Glu Phe Arg Arg Val Arg Gln Gly Tyr
405 410 415
Ser Gly Met Arg Asp Thr Leu Phe Glu Leu Ala Asn Leu Ile Arg Glu
420 425 430
Arg His His His Leu Ala His Tyr Arg Ser Pro Leu Arg Gln Asn Pro
435 440 445
Glu Ser Ser Leu Ser Thr Gly Gly Ala Tyr Ala Ala Asp
450 455 460
<210> 10
<211> 167
<212> PRT
<213> Искусственная последовательность
<220>
<223> Klebsiella sp. RCB570
<400> 10
Met Pro Pro Leu Asp Trp Leu Arg Arg Leu Trp Leu Leu Tyr His Ala
1. 5 10 15
Gly Lys Gly Ser Phe Pro Leu Arg Met Gly Leu Ser Pro Arg Asp Trp
20 25 30
Gln Ala Leu Arg Arg Arg Leu Gly Glu Val Glu Thr Pro Leu Asp Gly
35 40 45
Glu Thr Leu Thr Arg Arg Arg Leu Met Ala Glu Leu Asn Ala Thr Arg
50 55 60
Glu Glu Glu Arg Gln Gln Leu Gly Ala Trp Leu Ala Gly Trp Met Gln
65 70 75 80
Gln Asp Ala Gly Pro Met Ala Gln Ile Ile Ala Glu Val Ser Leu Ala
85 90 95
Phe Asn His Leu Trp Gln Asp Leu Gly Leu Ala Ser Arg Ala Glu Leu
100 105 110
Arg Leu Leu Met Ser Asp Cys Phe Pro Gln Leu Val Val Met Asn Glu
115 120 125
His Asn Met Arg Trp Lys Lys Phe Phe Tyr Arg Gln Arg Cys Leu Leu
130 135 140
Gln Gln Gly Glu Val Ile Cys Arg Ser Pro Ser Cys Asp Glu Cys Trp
145 150 155 160
Glu Arg Ser Ala Cys Phe Glu
165
<210> 11
<211> 400
<212> PRT
<213> Klebsiella oxytoca
<400> 11
Met Lys Gln Val Tyr Leu Asp Asn Asn Ala Thr Thr Arg Leu Asp Pro
1. 5 10 15
Met Val Leu Glu Ala Met Met Pro Phe Leu Thr Asp Phe Tyr Gly Asn
20 25 30
Pro Ser Ser Ile His Asp Phe Gly Ile Pro Ala Gln Ala Ala Leu Glu
35 40 45
Arg Ala His Gln Gln Ala Ala Ala Leu Leu Gly Ala Glu Tyr Pro Ser
50 55 60
Glu Ile Ile Phe Thr Ser Cys Ala Thr Glu Ala Thr Ala Thr Ala Ile
65 70 75 80
Ala Ser Ala Ile Ala Leu Leu Pro Glu Arg Arg Glu Ile Ile Thr Ser
85 90 95
Val Val Glu His Pro Ala Thr Leu Ala Ala Cys Glu His Met Glu Arg
100 105 110
Glu Gly Tyr Arg Ile His Arg Ile Ala Val Asp Gly Glu Gly Ala Leu
115 120 125
Asp Met Ala Gln Phe Arg Ala Ala Leu Ser Pro Arg Val Ala Leu Val
130 135 140
Ser Val Met Trp Ala Asn Asn Glu Thr Gly Val Leu Phe Pro Ile Gly
145 150 155 160
Glu Met Ala Glu Leu Ala His Glu Gln Gly Ala Leu Phe His Cys Asp
165 170 175
Ala Val Gln Val Val Gly Lys Ile Pro Ile Ala Val Gly Gln Thr Arg
180 185 190
Ile Asp Met Leu Ser Cys Ser Ala His Lys Phe His Gly Pro Lys Gly
195 200 205
Val Gly Cys Leu Tyr Leu Arg Arg Gly Thr Arg Phe Arg Pro Leu Leu
210 215 220
Arg Gly Gly His Gln Glu Tyr Gly Arg Arg Ala Gly Thr Glu Asn Ile
225 230 235 240
Cys Gly Ile Val Gly Met Gly Ala Ala Cys Glu Leu Ala Asn Ile His
245 250 255
Leu Pro Gly Met Thr His Ile Gly Gln Leu Arg Asn Arg Leu Glu His
260 265 270
Arg Leu Leu Ala Ser Val Pro Ser Val Met Val Met Gly Gly Gly Gln
275 280 285
Pro Ala Val Pro Gly Thr Val Asn Leu Ala Phe Glu Phe Ile Glu Gly
290 295 300
Glu Ala Ile Leu Leu Leu Leu Asn Gln Ala Gly Ile Ala Ala Ser Ser
305 310 315 320
Gly Ser Ala Cys Thr Ser Gly Ser Leu Glu Pro Ser His Val Met Arg
325 330 335
Ala Met Asn Ile Pro Tyr Thr Ala Ala His Gly Thr Ile Arg Phe Ser
340 345 350
Leu Ser Arg Tyr Thr Arg Glu Lys Glu Ile Asp Tyr Val Val Ala Thr
355 360 365
Leu Pro Pro Ile Ile Asp Arg Leu Arg Ala Leu Ser Pro Tyr Trp Gln
370 375 380
Asn Gly Lys Pro Arg Pro Ala Asp Ala Val Phe Thr Pro Val Tyr Gly
385 390 395 400
<210> 12
<211> 274
<212> PRT
<213> Klebsiella oxytoca
<400> 12
Met Trp Asn Tyr Ser Glu Lys Val Lys Asp His Phe Phe Asn Pro Arg
1. 5 10 15
Asn Ala Arg Val Val Asp Asn Ala Asn Ala Val Gly Asp Val Gly Ser
20 25 30
Leu Ser Cys Gly Asp Ala Leu Arg Leu Met Leu Arg Val Asp Pro Gln
35 40 45
Ser Glu Ile Ile Glu Glu Ala Gly Phe Gln Thr Phe Gly Cys Gly Ser
50 55 60
Ala Ile Ala Ser Ser Ser Ala Leu Thr Glu Leu Ile Ile Gly His Thr
65 70 75 80
Leu Ala Glu Ala Gly Gln Ile Thr Asn Gln Gln Ile Ala Asp Tyr Leu
85 90 95
Asp Gly Leu Pro Pro Glu Lys Met His Cys Ser Val Met Gly Gln Glu
100 105 110
Ala Leu Arg Ala Ala Ile Ala Asn Phe Arg Gly Glu Ser Leu Glu Glu
115 120 125
Glu His Asp Glu Gly Lys Leu Ile Cys Lys Cys Phe Gly Val Asp Glu
130 135 140
Gly His Ile Arg Arg Ala Val Gln Asn Asn Gly Leu Thr Thr Leu Ala
145 150 155 160
Glu Val Ile Asn Tyr Thr Lys Ala Gly Gly Gly Cys Thr Ser Cys His
165 170 175
Glu Lys Ile Glu Leu Ala Leu Ala Glu Ile Leu Ala Gln Gln Pro Gln
180 185 190
Thr Thr Pro Ala Val Ala Ser Gly Lys Asp Pro His Trp Gln Ser Val
195 200 205
Val Asp Thr Ile Ala Glu Leu Arg Pro His Ile Gln Ala Asp Gly Gly
210 215 220
Asp Met Ala Leu Leu Ser Val Thr Asn His Gln Val Thr Val Ser Leu
225 230 235 240
Ser Gly Ser Cys Ser Gly Cys Met Met Thr Asp Met Thr Leu Ala Trp
245 250 255
Leu Gln Gln Lys Leu Met Glu Arg Thr Gly Cys Tyr Met Glu Val Val
260 265 270
Ala Ala
<210> 13
<211> 381
<212> PRT
<213> Klebsiella oxytoca
<400> 13
Met Glu Arg Val Leu Ile Asn Asp Thr Thr Leu Arg Asp Gly Glu Gln
1. 5 10 15
Ser Pro Gly Val Ala Phe Arg Thr Ser Glu Lys Val Ala Ile Ala Glu
20 25 30
Ala Leu Tyr Ala Ala Gly Ile Thr Ala Met Glu Val Gly Thr Pro Ala
35 40 45
Met Gly Asp Glu Glu Ile Ala Arg Ile Gln Leu Val Arg Arg Gln Leu
50 55 60
Pro Asp Ala Thr Leu Met Thr Trp Cys Arg Met Asn Ala Leu Glu Ile
65 70 75 80
Arg Gln Ser Ala Asp Leu Gly Ile Asp Trp Val Asp Ile Ser Ile Pro
85 90 95
Ala Ser Asp Lys Leu Arg Gln Tyr Lys Leu Arg Glu Pro Leu Ala Val
100 105 110
Leu Leu Glu Arg Leu Ala Met Phe Ile His Leu Ala His Thr Leu Gly
115 120 125
Leu Lys Val Cys Ile Gly Cys Glu Asp Ala Ser Arg Ala Ser Gly Gln
130 135 140
Thr Leu Arg Ala Ile Ala Glu Val Ala Gln Asn Ala Pro Ala Ala Arg
145 150 155 160
Leu Arg Tyr Ala Asp Thr Val Gly Leu Leu Asp Pro Phe Thr Thr Ala
165 170 175
Ala Gln Ile Ser Ala Leu Arg Asp Val Trp Ser Gly Glu Ile Glu Met
180 185 190
His Ala His Asn Asp Leu Gly Met Ala Thr Ala Asn Thr Leu Ala Ala
195 200 205
Val Ser Ala Gly Ala Thr Ser Val Asn Thr Thr Val Leu Gly Leu Gly
210 215 220
Glu Arg Ala Gly Asn Ala Ala Ala Trp Lys Pro Ser Ala Leu Gly Leu
225 230 235 240
Glu Arg Cys Leu Gly Val Glu Thr Gly Val His Phe Ser Ala Leu Pro
245 250 255
Ala Leu Cys Gln Arg Val Ala Glu Ala Ala Gln Arg Ala Ile Asp Pro
260 265 270
Gln Gln Pro Leu Val Gly Glu Leu Val Phe Thr His Glu Ser Gly Val
275 280 285
His Val Ala Ala Leu Leu Arg Asp Ser Glu Ser Tyr Gln Ser Ile Ala
290 295 300
Pro Ser Leu Met Gly Arg Ser Tyr Arg Leu Val Leu Gly Lys His Ser
305 310 315 320
Gly Arg Gln Ala Val Asn Gly Val Phe Asp Gln Met Gly Tyr His Leu
325 330 335
Asn Ala Ala Gln Ile Asn Gln Leu Leu Pro Ala Ile Arg Arg Phe Ala
340 345 350
Glu Asn Trp Lys Arg Ser Pro Lys Asp Tyr Glu Leu Val Ala Ile Tyr
355 360 365
Asp Glu Leu Cys Gly Glu Ser Ala Leu Arg Ala Arg Gly
370 375 380
<210> 14
<211> 156
<212> PRT
<213> Klebsiella oxytoca
<400> 14
Met Pro Pro Ile Asn Arg Gln Phe Asp Met Val His Ser Asp Glu Trp
1. 5 10 15
Ser Met Lys Val Ala Phe Ala Ser Ser Asp Tyr Arg His Val Asp Gln
20 25 30
His Phe Gly Ala Thr Pro Arg Leu Val Val Tyr Gly Val Lys Ala Asp
35 40 45
Arg Val Thr Leu Ile Arg Val Val Asp Phe Ser Val Glu Asn Gly His
50 55 60
Gln Thr Glu Lys Ile Ala Arg Arg Ile His Ala Leu Glu Asp Cys Val
65 70 75 80
Thr Leu Phe Cys Val Ala Ile Gly Asp Ala Val Phe Arg Gln Leu Leu
85 90 95
Gln Val Gly Val Arg Ala Glu Arg Val Pro Ala Asp Thr Thr Ile Val
100 105 110
Gly Leu Leu Gln Glu Ile Gln Leu Tyr Trp Tyr Asp Lys Gly Gln Arg
115 120 125
Lys Asn Gln Arg Gln Arg Asp Pro Glu Arg Phe Thr Arg Leu Leu Gln
130 135 140
Glu Gln Glu Trp His Gly Asp Pro Asp Pro Arg Arg
145 150 155
<210> 15
<211> 220
<212> PRT
<213> Klebsiella oxytoca
<400> 15
Met Ser Asp Asn Asp Thr Leu Phe Trp Arg Met Leu Ala Leu Phe Gln
1. 5 10 15
Ser Leu Pro Asp Leu Gln Pro Ala Gln Ile Val Asp Trp Leu Ala Gln
20 25 30
Glu Ser Gly Glu Thr Leu Thr Pro Glu Arg Leu Ala Thr Leu Thr Gln
35 40 45
Pro Gln Leu Ala Ala Ser Phe Pro Ser Ala Thr Ala Val Met Ser Pro
50 55 60
Ala Arg Trp Ser Arg Val Met Ala Ser Leu Gln Gly Ala Leu Pro Ala
65 70 75 80
His Leu Arg Ile Val Arg Pro Ala Gln Arg Thr Pro Gln Leu Leu Ala
85 90 95
Ala Phe Cys Ser Gln Asp Gly Leu Val Ile Asn Gly His Phe Gly Gln
100 105 110
Gly Arg Leu Phe Phe Ile Tyr Ala Phe Asp Glu Gln Gly Gly Trp Leu
115 120 125
Tyr Asp Leu Arg Arg Tyr Pro Ser Ala Pro His Gln Gln Glu Ala Asn
130 135 140
Glu Val Arg Ala Arg Leu Ile Glu Asp Cys Gln Leu Leu Phe Cys Gln
145 150 155 160
Glu Ile Gly Gly Pro Ala Ala Ala Arg Pro Ile Arg His Arg Ile His
165 170 175
Pro Met Lys Ala Gln Pro Gly Thr Thr Ile Gln Ala Gln Cys Glu Ala
180 185 190
Ile Asn Thr Leu Leu Ala Gly Arg Leu Pro Pro Trp Leu Ala Lys Arg
195 200 205
Leu Asn Arg Asp Asn Pro Leu Glu Glu Arg Val Phe
210 215 220
<210> 16
<211> 148
<212> PRT
<213> Klebsiella oxytoca
<400> 16
Met Arg Pro Lys Phe Thr Phe Ser Glu Glu Val Arg Val Val Arg Ala
1. 5 10 15
Ile Arg Asn Asp Gly Thr Val Ala Gly Phe Ala Pro Gly Ala Leu Leu
20 25 30
Val Arg Arg Gly Ser Thr Gly Phe Val Arg Asp Trp Gly Val Phe Leu
35 40 45
Gln Asp Gln Ile Ile Tyr Gln Ile His Phe Pro Glu Thr Asp Arg Ile
50 55 60
Ile Gly Cys Arg Glu Gln Glu Leu Ile Pro Ile Thr Gln Pro Trp Leu
65 70 75 80
Ala Gly Asn Leu Gln Tyr Arg Asp Ser Val Thr Cys Gln Met Ala Leu
85 90 95
Ala Val Asn Gly Asp Val Val Val Ser Ala Gly Gln Arg Gly Arg Val
100 105 110
Glu Ala Thr Asp Arg Gly Glu Leu Gly Asp Ser Tyr Thr Val Asp Phe
115 120 125
Ser Gly Arg Trp Phe Arg Val Pro Val Gln Ala Ile Ala Leu Ile Glu
130 135 140
Glu Arg Glu Glu
145
<210> 17
<211> 88
<212> PRT
<213> Klebsiella oxytoca
<400> 17
Met Met Glu Trp Phe Tyr Gln Ile Pro Gly Val Asp Glu Leu Arg Ser
1. 5 10 15
Ala Glu Ser Phe Phe Gln Phe Phe Ala Val Pro Tyr Gln Pro Glu Leu
20 25 30
Leu Gly Arg Cys Ser Leu Pro Val Leu Ala Thr Phe His Arg Lys Leu
35 40 45
Arg Ala Glu Val Pro Leu Gln Asn Arg Leu Glu Asp Asn Asp Arg Ala
50 55 60
Pro Trp Leu Leu Ala Arg Arg Leu Leu Ala Glu Ser Tyr Gln Gln Gln
65 70 75 80
Phe Gln Glu Ser Gly Thr Gly Gly
85
<210> 18
<211> 483
<212> PRT
<213> Klebsiella oxytoca
<400> 18
Met Met Thr Asn Ala Thr Gly Glu Arg Asn Leu Ala Leu Ile Gln Glu
1. 5 10 15
Val Leu Glu Val Phe Pro Glu Thr Ala Arg Lys Glu Arg Arg Lys His
20 25 30
Met Met Val Ser Asp Pro Glu Met Glu Ser Val Gly Lys Cys Ile Ile
35 40 45
Ser Asn Arg Lys Ser Gln Pro Gly Val Met Thr Val Arg Gly Cys Ala
50 55 60
Tyr Ala Gly Ser Lys Gly Val Val Phe Gly Pro Ile Lys Asp Met Ala
65 70 75 80
His Ile Ser His Gly Pro Val Gly Cys Gly Gln Tyr Ser Arg Ala Gly
85 90 95
Arg Arg Asn Tyr Tyr Thr Gly Val Ser Gly Val Asp Ser Phe Gly Thr
100 105 110
Leu Asn Phe Thr Ser Asp Phe Gln Glu Arg Asp Ile Val Phe Gly Gly
115 120 125
Asp Lys Lys Leu Ser Lys Leu Ile Glu Glu Met Glu Leu Leu Phe Pro
130 135 140
Leu Thr Lys Gly Ile Thr Ile Gln Ser Glu Cys Pro Val Gly Leu Ile
145 150 155 160
Gly Asp Asp Ile Ser Ala Val Ala Asn Ala Ser Ser Lys Ala Leu Asp
165 170 175
Lys Pro Val Ile Pro Val Arg Cys Glu Gly Phe Arg Gly Val Ser Gln
180 185 190
Ser Leu Gly His His Ile Ala Asn Asp Val Val Arg Asp Trp Ile Leu
195 200 205
Asn Asn Arg Glu Gly Gln Pro Phe Glu Thr Thr Pro Tyr Asp Val Ala
210 215 220
Ile Ile Gly Asp Tyr Asn Ile Gly Gly Asp Ala Trp Ala Ser Arg Ile
225 230 235 240
Leu Leu Glu Glu Met Gly Leu Arg Val Val Ala Gln Trp Ser Gly Asp
245 250 255
Gly Thr Leu Val Glu Met Glu Asn Thr Pro Phe Val Lys Leu Asn Leu
260 265 270
Val His Cys Tyr Arg Ser Met Asn Tyr Ile Ala Arg His Met Glu Glu
275 280 285
Lys His Gln Ile Pro Trp Met Glu Tyr Asn Phe Phe Gly Pro Thr Lys
290 295 300
Ile Ala Glu Ser Leu Arg Lys Ile Ala Asp Gln Phe Asp Asp Thr Ile
305 310 315 320
Arg Ala Asn Ala Glu Ala Val Ile Ala Arg Tyr Glu Gly Gln Met Ala
325 330 335
Ala Ile Ile Ala Lys Tyr Arg Pro Arg Leu Glu Gly Arg Lys Val Leu
340 345 350
Leu Tyr Met Gly Gly Leu Arg Pro Arg His Val Ile Gly Ala Tyr Glu
355 360 365
Asp Leu Gly Met Glu Ile Ile Ala Ala Gly Tyr Glu Phe Ala His Asn
370 375 380
Asp Asp Tyr Asp Arg Thr Leu Pro Asp Leu Lys Glu Gly Thr Leu Leu
385 390 395 400
Phe Asp Asp Ala Ser Ser Tyr Glu Leu Glu Ala Phe Val Lys Ala Leu
405 410 415
Lys Pro Asp Leu Ile Gly Ser Gly Ile Lys Glu Lys Tyr Ile Phe Gln
420 425 430
Lys Met Gly Val Pro Phe Arg Gln Met His Ser Trp Asp Tyr Ser Gly
435 440 445
Pro Tyr His Gly Tyr Asp Gly Phe Ala Ile Phe Ala Arg Asp Met Asp
450 455 460
Met Thr Leu Asn Asn Pro Ala Trp Asn Glu Leu Thr Ala Pro Trp Leu
465 470 475 480
Lys Ser Ala
<210> 19
<211> 400
<212> PRT
<213> Klebsiella oxytoca
<400> 19
Met Lys Gln Val Tyr Leu Asp Asn Asn Ala Thr Thr Arg Leu Asp Pro
1. 5 10 15
Met Val Leu Glu Ala Met Met Pro Phe Leu Thr Asp Phe Tyr Gly Asn
20 25 30
Pro Ser Ser Ile His Asp Phe Gly Ile Pro Ala Gln Ala Ala Leu Glu
35 40 45
Arg Ala His Gln Gln Ala Ala Ala Leu Leu Gly Ala Glu Tyr Pro Ser
50 55 60
Glu Ile Ile Phe Thr Ser Cys Ala Thr Glu Ala Thr Ala Thr Ala Ile
65 70 75 80
Ala Ser Ala Ile Ala Leu Leu Pro Glu Arg Arg Glu Ile Ile Thr Ser
85 90 95
Val Val Glu His Pro Ala Thr Leu Ala Ala Cys Glu His Leu Glu Arg
100 105 110
Gln Gly Tyr Arg Ile His Arg Ile Ala Val Asp Ser Glu Gly Ala Leu
115 120 125
Asp Met Ala Gln Phe Arg Ala Ala Leu Ser Pro Arg Val Ala Leu Val
130 135 140
Ser Val Met Trp Ala Asn Asn Glu Thr Gly Val Leu Phe Pro Ile Gly
145 150 155 160
Glu Met Ala Glu Leu Ala His Glu Gln Gly Ala Leu Phe His Cys Asp
165 170 175
Ala Val Gln Val Val Gly Lys Ile Pro Ile Ala Val Gly Gln Thr Arg
180 185 190
Ile Asp Met Leu Ser Cys Ser Ala His Lys Phe His Gly Pro Lys Gly
195 200 205
Val Gly Cys Leu Tyr Leu Arg Arg Gly Thr Arg Phe Arg Pro Leu Leu
210 215 220
Arg Gly Gly His Gln Glu Tyr Gly Arg Arg Ala Gly Thr Glu Asn Ile
225 230 235 240
Cys Gly Ile Val Gly Met Gly Ala Ala Cys Glu Leu Ala Asn Ile His
245 250 255
Leu Pro Gly Met Thr His Ile Gly Gln Leu Arg Asn Arg Leu Glu His
260 265 270
Arg Leu Leu Ala Ser Val Pro Ser Val Met Val Met Gly Gly Gly Gln
275 280 285
Pro Arg Val Pro Gly Thr Val Asn Leu Ala Phe Glu Phe Ile Glu Gly
290 295 300
Glu Ala Ile Leu Leu Leu Leu Asn Gln Ala Gly Ile Ala Ala Ser Ser
305 310 315 320
Gly Ser Ala Cys Thr Ser Gly Ser Leu Glu Pro Ser His Val Met Arg
325 330 335
Ala Met Asn Ile Pro Tyr Thr Ala Ala His Gly Thr Ile Arg Phe Ser
340 345 350
Leu Ser Arg Tyr Thr Arg Glu Lys Glu Ile Asp Tyr Val Val Ala Thr
355 360 365
Leu Pro Pro Ile Ile Asp Arg Leu Arg Ala Leu Ser Pro Tyr Trp Gln
370 375 380
Asn Gly Lys Pro Arg Pro Ala Asp Ala Val Phe Thr Pro Val Tyr Gly
385 390 395 400
<210> 20
<211> 80
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность N-концевого продолжения, содержащая
pFA MTP (аминокислоты 1-77) и триплетные аминокислоты GAP
(78-80). Расщепление с помощью MPP имеет место между остатками
аминокислот 42 и 43
<400> 20
Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro Ser
1. 5 10 15
Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser Asp
20 25 30
Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val Val
35 40 45
Arg Asn Arg Met Lys Ser Val Lys Asn Ile Gln Lys Ile Thr Lys Ala
50 55 60
Met Lys Met Val Ala Ala Ser Lys Leu Arg Ala Val Gln Gly Ala Pro
65 70 75 80
<210> 21
<211> 54
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность MTP-FA?51 полипептида с
дополнительным N-концевым Met и C-концевым GG. Расщепление с
помощью MPP имеет место между остатками аминокислот 43 и 44.
<400> 21
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly
50
<210> 22
<211> 9
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность полипептида FAv-scar9.
<400> 22
Ile Ser Thr Gln Val Val Arg Asn Arg
1. 5
<210> 23
<211> 389
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-FA
77::NifH::HA, кодированная pRA10. Аминокислоты 1-77 соответствуют
MTP-FA 77, аминокислоты 78-80 представляют собой GAP, аминокислоты
81-372 представляли собой аминокислоты NifH K.oxytoca,а аминокислоты
373-389 включают HA
<400> 23
Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro Ser
1. 5 10 15
Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser Asp
20 25 30
Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val Val
35 40 45
Arg Asn Arg Met Lys Ser Val Lys Asn Ile Gln Lys Ile Thr Lys Ala
50 55 60
Met Lys Met Val Ala Ala Ser Lys Leu Arg Ala Val Gln Gly Ala Pro
65 70 75 80
Thr Met Arg Gln Cys Ala Ile Tyr Gly Lys Gly Gly Ile Gly Lys Ser
85 90 95
Thr Thr Thr Gln Asn Leu Val Ala Ala Leu Ala Glu Met Gly Lys Lys
100 105 110
Val Met Ile Val Gly Cys Asp Pro Lys Ala Asp Ser Thr Arg Leu Ile
115 120 125
Leu His Ala Lys Ala Gln Asn Thr Ile Met Glu Met Ala Ala Glu Val
130 135 140
Gly Ser Val Glu Asp Leu Glu Leu Glu Asp Val Leu Gln Ile Gly Tyr
145 150 155 160
Gly Asp Val Arg Cys Ala Glu Ser Gly Gly Pro Glu Pro Gly Val Gly
165 170 175
Cys Ala Gly Arg Gly Val Ile Thr Ala Ile Asn Phe Leu Glu Glu Glu
180 185 190
Gly Ala Tyr Glu Asp Asp Leu Asp Phe Val Phe Tyr Asp Val Leu Gly
195 200 205
Asp Val Val Cys Gly Gly Phe Ala Met Pro Ile Arg Glu Asn Lys Ala
210 215 220
Gln Glu Ile Tyr Ile Val Cys Ser Gly Glu Met Met Ala Met Tyr Ala
225 230 235 240
Ala Asn Asn Ile Ser Lys Gly Ile Val Lys Tyr Ala Lys Ser Gly Lys
245 250 255
Val Arg Leu Gly Gly Leu Ile Cys Asn Ser Arg Gln Thr Asp Arg Glu
260 265 270
Asp Glu Leu Ile Ile Ala Leu Ala Glu Lys Leu Gly Thr Gln Met Ile
275 280 285
His Phe Val Pro Arg Asp Asn Ile Val Gln Arg Ala Glu Ile Arg Arg
290 295 300
Met Thr Val Ile Glu Tyr Asp Pro Ala Cys Lys Gln Ala Asn Glu Tyr
305 310 315 320
Arg Thr Leu Ala Gln Lys Ile Val Asn Asn Thr Met Lys Val Val Pro
325 330 335
Thr Pro Cys Thr Met Asp Glu Leu Glu Ser Leu Leu Met Glu Phe Gly
340 345 350
Ile Met Glu Glu Glu Asp Thr Ser Ile Ile Gly Lys Thr Ala Ala Glu
355 360 365
Glu Asn Ala Ala Ala Gly Gly Gly Gly Gly Tyr Pro Tyr Asp Val Pro
370 375 380
Asp Tyr Ala Pro Gly
385
<210> 24
<211> 363
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-FA
51::NifH::HA, кодированная pRA34. Аминокислоты 1-51 соответствуют
MTP-FA 51, аминокислоты 52-54 представляют собой GAP, аминокислоты
55-346 представляли собой аминокислоты NifH K.oxytoca, а аминокислоты
347-363 включают HA
<400> 24
Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro Ser
1. 5 10 15
Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser Asp
20 25 30
Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val Val
35 40 45
Arg Asn Arg Gly Ala Pro Thr Met Arg Gln Cys Ala Ile Tyr Gly Lys
50 55 60
Gly Gly Ile Gly Lys Ser Thr Thr Thr Gln Asn Leu Val Ala Ala Leu
65 70 75 80
Ala Glu Met Gly Lys Lys Val Met Ile Val Gly Cys Asp Pro Lys Ala
85 90 95
Asp Ser Thr Arg Leu Ile Leu His Ala Lys Ala Gln Asn Thr Ile Met
100 105 110
Glu Met Ala Ala Glu Val Gly Ser Val Glu Asp Leu Glu Leu Glu Asp
115 120 125
Val Leu Gln Ile Gly Tyr Gly Asp Val Arg Cys Ala Glu Ser Gly Gly
130 135 140
Pro Glu Pro Gly Val Gly Cys Ala Gly Arg Gly Val Ile Thr Ala Ile
145 150 155 160
Asn Phe Leu Glu Glu Glu Gly Ala Tyr Glu Asp Asp Leu Asp Phe Val
165 170 175
Phe Tyr Asp Val Leu Gly Asp Val Val Cys Gly Gly Phe Ala Met Pro
180 185 190
Ile Arg Glu Asn Lys Ala Gln Glu Ile Tyr Ile Val Cys Ser Gly Glu
195 200 205
Met Met Ala Met Tyr Ala Ala Asn Asn Ile Ser Lys Gly Ile Val Lys
210 215 220
Tyr Ala Lys Ser Gly Lys Val Arg Leu Gly Gly Leu Ile Cys Asn Ser
225 230 235 240
Arg Gln Thr Asp Arg Glu Asp Glu Leu Ile Ile Ala Leu Ala Glu Lys
245 250 255
Leu Gly Thr Gln Met Ile His Phe Val Pro Arg Asp Asn Ile Val Gln
260 265 270
Arg Ala Glu Ile Arg Arg Met Thr Val Ile Glu Tyr Asp Pro Ala Cys
275 280 285
Lys Gln Ala Asn Glu Tyr Arg Thr Leu Ala Gln Lys Ile Val Asn Asn
290 295 300
Thr Met Lys Val Val Pro Thr Pro Cys Thr Met Asp Glu Leu Glu Ser
305 310 315 320
Leu Leu Met Glu Phe Gly Ile Met Glu Glu Glu Asp Thr Ser Ile Ile
325 330 335
Gly Lys Thr Ala Ala Glu Glu Asn Ala Ala Ala Gly Gly Gly Gly Gly
340 345 350
Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Pro Gly
355 360
<210> 25
<211> 358
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-
FAv51::NifH::HA, кодированная SN18. Аминокислоты 1-54 соответствуют
MTP-FA 51 с GG, аминокислоты 55-347 представляли собой аминокислоты
NifH K.oxytoca, а аминокислоты 348-358 включают эпитоп HA
<400> 25
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly Met Thr Met Arg Gln Cys Ala Ile Tyr Gly
50 55 60
Lys Gly Gly Ile Gly Lys Ser Thr Thr Thr Gln Asn Leu Val Ala Ala
65 70 75 80
Leu Ala Glu Met Gly Lys Lys Val Met Ile Val Gly Cys Asp Pro Lys
85 90 95
Ala Asp Ser Thr Arg Leu Ile Leu His Ala Lys Ala Gln Asn Thr Ile
100 105 110
Met Glu Met Ala Ala Glu Val Gly Ser Val Glu Asp Leu Glu Leu Glu
115 120 125
Asp Val Leu Gln Ile Gly Tyr Gly Asp Val Arg Cys Ala Glu Ser Gly
130 135 140
Gly Pro Glu Pro Gly Val Gly Cys Ala Gly Arg Gly Val Ile Thr Ala
145 150 155 160
Ile Asn Phe Leu Glu Glu Glu Gly Ala Tyr Glu Asp Asp Leu Asp Phe
165 170 175
Val Phe Tyr Asp Val Leu Gly Asp Val Val Cys Gly Gly Phe Ala Met
180 185 190
Pro Ile Arg Glu Asn Lys Ala Gln Glu Ile Tyr Ile Val Cys Ser Gly
195 200 205
Glu Met Met Ala Met Tyr Ala Ala Asn Asn Ile Ser Lys Gly Ile Val
210 215 220
Lys Tyr Ala Lys Ser Gly Lys Val Arg Leu Gly Gly Leu Ile Cys Asn
225 230 235 240
Ser Arg Gln Thr Asp Arg Glu Asp Glu Leu Ile Ile Ala Leu Ala Glu
245 250 255
Lys Leu Gly Thr Gln Met Ile His Phe Val Pro Arg Asp Asn Ile Val
260 265 270
Gln Arg Ala Glu Ile Arg Arg Met Thr Val Ile Glu Tyr Asp Pro Ala
275 280 285
Cys Lys Gln Ala Asn Glu Tyr Arg Thr Leu Ala Gln Lys Ile Val Asn
290 295 300
Asn Thr Met Lys Val Val Pro Thr Pro Cys Thr Met Asp Glu Leu Glu
305 310 315 320
Ser Leu Leu Met Glu Phe Gly Ile Met Glu Glu Glu Asp Thr Ser Ile
325 330 335
Ile Gly Lys Thr Ala Ala Glu Glu Asn Ala Ala Gly Gly Tyr Pro Tyr
340 345 350
Asp Val Pro Asp Tyr Ala
355
<210> 26
<211> 371
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-
FAv51::HA::NifH, кодированная SN29. Аминокислоты 1-53 соответствуют
MTP-FA 51 с GG, аминокислоты 54-64 включают эпитоп HA, аминокислоты
65-357 соответствуют NifH K.oxytoca и аминокислотам 358-371
<400> 26
Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro Ser
1. 5 10 15
Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser Asp
20 25 30
Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val Val
35 40 45
Arg Asn Arg Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Gly Gly
50 55 60
Met Thr Met Arg Gln Cys Ala Ile Tyr Gly Lys Gly Gly Ile Gly Lys
65 70 75 80
Ser Thr Thr Thr Gln Asn Leu Val Ala Ala Leu Ala Glu Met Gly Lys
85 90 95
Lys Val Met Ile Val Gly Cys Asp Pro Lys Ala Asp Ser Thr Arg Leu
100 105 110
Ile Leu His Ala Lys Ala Gln Asn Thr Ile Met Glu Met Ala Ala Glu
115 120 125
Val Gly Ser Val Glu Asp Leu Glu Leu Glu Asp Val Leu Gln Ile Gly
130 135 140
Tyr Gly Asp Val Arg Cys Ala Glu Ser Gly Gly Pro Glu Pro Gly Val
145 150 155 160
Gly Cys Ala Gly Arg Gly Val Ile Thr Ala Ile Asn Phe Leu Glu Glu
165 170 175
Glu Gly Ala Tyr Glu Asp Asp Leu Asp Phe Val Phe Tyr Asp Val Leu
180 185 190
Gly Asp Val Val Cys Gly Gly Phe Ala Met Pro Ile Arg Glu Asn Lys
195 200 205
Ala Gln Glu Ile Tyr Ile Val Cys Ser Gly Glu Met Met Ala Met Tyr
210 215 220
Ala Ala Asn Asn Ile Ser Lys Gly Ile Val Lys Tyr Ala Lys Ser Gly
225 230 235 240
Lys Val Arg Leu Gly Gly Leu Ile Cys Asn Ser Arg Gln Thr Asp Arg
245 250 255
Glu Asp Glu Leu Ile Ile Ala Leu Ala Glu Lys Leu Gly Thr Gln Met
260 265 270
Ile His Phe Val Pro Arg Asp Asn Ile Val Gln Arg Ala Glu Ile Arg
275 280 285
Arg Met Thr Val Ile Glu Tyr Asp Pro Ala Cys Lys Gln Ala Asn Glu
290 295 300
Tyr Arg Thr Leu Ala Gln Lys Ile Val Asn Asn Thr Met Lys Val Val
305 310 315 320
Pro Thr Pro Cys Thr Met Asp Glu Leu Glu Ser Leu Leu Met Glu Phe
325 330 335
Gly Ile Met Glu Glu Glu Asp Thr Ser Ile Ile Gly Lys Thr Ala Ala
340 345 350
Glu Glu Asn Ala Ala Ala Ser Leu Ala Arg Val Asp Arg Gln Ala Arg
355 360 365
Val Ser Pro
370
<210> 27
<211> 9
<212> PRT
<213> Искусственная последовательность
<220>
<223> последовательность 6xHis использовалась вместо MTP, с N-концевым Met и
C-концевым GG
<400> 27
Met His His His His His His Gly Gly
1. 5
<210> 28
<211> 33
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность CPN60 MTP
<400> 28
Met Tyr Arg Phe Ala Ser Asn Leu Ala Ser Lys Ala Arg Ile Ala Gln
1. 5 10 15
Asn Ala Arg Gln Val Ser Ser Arg Met Ser Trp Ser Arg Asn Tyr Gly
20 25 30
Gly
<210> 29
<211> 31
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность CPN60 / без линкера GG MTP
<400> 29
Met Tyr Arg Phe Ala Ser Asn Leu Ala Ser Lys Ala Arg Ile Ala Gln
1. 5 10 15
Asn Ala Arg Gln Val Ser Ser Arg Met Ser Trp Ser Arg Asn Tyr
20 25 30
<210> 30
<211> 31
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность супероксид-дисмутазы (SOD) MTP
<400> 30
Met Ala Ile Arg Cys Val Ala Ser Arg Lys Thr Leu Ala Gly Leu Lys
1. 5 10 15
Glu Thr Ser Ser Arg Leu Leu Arg Ile Arg Gly Ile Gln Gly Gly
20 25 30
<210> 31
<211> 60
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность удвоенной супероксид-дисмутазы
(2SOD)MTP
<400> 31
Met Ala Ile Arg Cys Val Ala Ser Arg Lys Thr Leu Ala Gly Leu Lys
1. 5 10 15
Glu Thr Ser Ser Arg Leu Leu Arg Ile Arg Gly Ile Gln Met Ala Ile
20 25 30
Arg Cys Val Ala Ser Arg Lys Thr Leu Ala Gly Leu Lys Glu Thr Ser
35 40 45
Ser Arg Leu Leu Arg Ile Arg Gly Ile Gln Gly Gly
50 55 60
<210> 32
<211> 29
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность супероксид-дисмутазы,
модифицированная удвоенная (SODmod) MTP
<400> 32
Met Ala Ile Arg Cys Val Ala Ser Arg Lys Thr Leu Ala Gly Leu Lys
1. 5 10 15
Glu Thr Ser Ser Arg Leu Leu Arg Ile Arg Gly Gly Gly
20 25
<210> 33
<211> 56
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность супероксид-дисмутазы,
модифицированная удвоенная (2SODmod) MTP
<400> 33
Met Ala Ile Arg Cys Val Ala Ser Arg Lys Thr Leu Ala Gly Leu Lys
1. 5 10 15
Glu Thr Ser Ser Arg Leu Leu Arg Ile Arg Gly Met Ala Ile Arg Cys
20 25 30
Val Ala Ser Arg Lys Thr Leu Ala Gly Leu Lys Glu Thr Ser Ser Arg
35 40 45
Leu Leu Arg Ile Arg Gly Gly Gly
50 55
<210> 34
<211> 34
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность L29 MTP (At1G07830)
<400> 34
Met Phe Leu Thr Arg Phe Val Gly Arg Arg Phe Leu Ala Ala Ala Ser
1. 5 10 15
Ala Arg Ser Glu Ser Thr Thr Ala Ala Ala Ala Ala Ser Thr Ile Arg
20 25 30
Gly Gly
<210> 35
<211> 70
<212> PRT
<213> Нейроспора красса
<400> 35
Met Ala Ser Thr Arg Val Leu Ala Ser Arg Leu Ala Ser Gln Met Ala
1. 5 10 15
Ala Ser Ala Lys Val Ala Arg Pro Ala Val Arg Val Ala Gln Val Ser
20 25 30
Lys Arg Thr Ile Gln Thr Gly Ser Pro Leu Gln Thr Leu Lys Arg Thr
35 40 45
Gln Met Thr Ser Ile Val Asn Ala Thr Thr Arg Gln Ala Phe Gln Lys
50 55 60
Arg Ala Tyr Ser Gly Gly
65 70
<210> 36
<211> 53
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность гамма-субъединицы gATPase (FAv51)
MTP, без дополнительной N-концевой Met. SEQ ID NO:21 имеет
дополнительную N-концевую Met. Расщепление с помощью MPP имеет место
между аминокислотными остатками 42 и 43.
<400> 36
Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro Ser
1. 5 10 15
Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser Asp
20 25 30
Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val Val
35 40 45
Arg Asn Arg Gly Gly
50
<210> 37
<211> 61
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность CoxIV TwinStrep (ABM97483) MTP
<400> 37
Met Leu Ser Leu Arg Gln Ser Ile Arg Phe Phe Lys Pro Ala Thr Arg
1. 5 10 15
Thr Leu Cys Ser Ser Arg Tyr Leu Leu Gln Gln Lys Pro Ser Ala Trp
20 25 30
Ser His Pro Gln Phe Glu Lys Gly Gly Gly Ser Gly Gly Gly Ser Gly
35 40 45
Gly Ser Ala Trp Ser His Pro Gln Phe Glu Lys Gly Gly
50 55 60
<210> 38
<211> 43
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность CoxIV 10xHis (ABM97483) MTP
<400> 38
Met Leu Ser Leu Arg Gln Ser Ile Arg Phe Phe Lys Pro Ala Thr Arg
1. 5 10 15
Thr Leu Cys Ser Ser Arg Tyr Leu Leu Gln Gln Lys Pro Gly Gly His
20 25 30
His His His His His His His His His Gly Gly
35 40
<210> 39
<211> 4
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность предсказанной scar для супероксид-
дисмутазы (SOD) MTP с GG
<400> 39
Ile Gln Gly Gly
1
<210> 40
<211> 15
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность предсказанной scar для двойной
супероксид-дисмутазы, (2SOD) MTP с GG
<400> 40
Glu Ser Thr Thr Ala Ala Ala Ala Ala Ser Thr Ile Arg Gly Gly
1. 5 10 15
<210> 41
<211> 4
<212> PRT
<213> Нейроспора красса
<400> 41
Tyr Ser Gly Gly
1
<210> 42
<211> 11
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность предсказанной scar для гамма-
субъединицы gATPase (FA?51) MTP с GG
<400> 42
Ile Ser Thr Gln Val Val Arg Asn Arg Gly Gly
1. 5 10
<210> 43
<211> 36
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность предсказанной scar для CoxIV
TwinStrep MTP с GG
<400> 43
Gln Gln Lys Pro Ser Ala Trp Ser His Pro Gln Phe Glu Lys Gly Gly
1. 5 10 15
Gly Ser Gly Gly Gly Ser Gly Gly Ser Ala Trp Ser His Pro Gln Phe
20 25 30
Glu Lys Gly Gly
35
<210> 44
<211> 18
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность предсказанной scar для CoxIV
10xHis MTP с GG
<400> 44
Gln Gln Lys Pro Gly Gly His His His His His His His His His His
1. 5 10 15
Gly Gly
<210> 45
<211> 40
<212> DNA
<213> Искусственная последовательность
<220>
<223> Олигонуклеотидный праймер MIT_V2.1_SbfInifH_FW2
<400> 45
aacctgcagg tgacgtctaa gaaaaggaat attcagcaat 40
<210> 46
<211> 39
<212> DNA
<213> Искусственная последовательность
<220>
<223> Олигонуклеотидный праймер MIT_V2.1_SbfInifJ_RV2
<400> 46
aacctgcagg gctaactaac taaccacgga caaaaaacc 39
<210> 47
<211> 35
<212> DNA
<213> Искусственная последовательность
<220>
<223> Олигонуклеотидный праймер MIT_V2.1_SbfInifB_FW
<400> 47
aacctgcagg tactctaacc ccatcggccg tctta 35
<210> 48
<211> 35
<212> DNA
<213> Искусственная последовательность
<220>
<223> Олигонуклеотидный праймер MIT_V2.1_SbfIori_RV
<400> 48
aacctgcagg tacgtagcaa tcaactcact ggctc 35
<210> 49
<211> 9
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность mscar9 из MTP-FAv51, с заменой
N-концевого остатка Ile на Met для инициации трансляции
<400> 49
Met Ser Thr Gln Val Val Arg Asn Arg
1. 5
<210> 50
<211> 6
<212> PRT
<213> Искусственная последовательность
<220>
<223> Триптический пептид
<400> 50
Ser Thr Gln Val Val Arg
1. 5
<210> 51
<211> 9
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность MTP-FAv9 scar, без N-концевой Met и
с C-концевой Met
<400> 51
Ser Thr Gln Val Val Arg Asn Arg Met
1. 5
<210> 52
<211> 47
<212> DNA
<213> Искусственная последовательность
<220>
<223> Олигонуклеотидный праймер
<400> 52
atgtcaactc aagtggtgcg taaccgcatg acctcttgtt cgtcgtt 47
<210> 53
<211> 40
<212> DNA
<213> Искусственная последовательность
<220>
<223> Олигонуклеотидный праймер
<400> 53
tttagccctc ctatgattga tttgatgtat tacagagagg 40
<210> 54
<211> 52
<212> DNA
<213> Искусственная последовательность
<220>
<223> Олигонуклеотидный праймер
<400> 54
ggttacgcac cacttgagtt gacattttag ccctcctatg attgatttga tg 52
<210> 55
<211> 7
<212> PRT
<213> Искусственная последовательность
<220>
<223> Триптический пептид
<400> 55
Met Ser Thr Gln Val Val Arg
1. 5
<210> 56
<211> 8
<212> PRT
<213> Искусственная последовательность
<220>
<223> Триптический пептид
<400> 56
Thr Gln Val Val Arg Asn Arg
1. 5
<210> 57
<211> 599
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-FA 77::NifK,
(pRA25), без любого C-концевого продолжения. Аминокислоты 1-77
соответствуют MTP-FA 77, аминокислоты 78-80 представляют собой GAP,
а аминокислоты 81-599 соответствуют NifK K. oxytoca без инициатора
<400> 57
Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro Ser
1. 5 10 15
Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser Asp
20 25 30
Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val Val
35 40 45
Arg Asn Arg Met Lys Ser Val Lys Asn Ile Gln Lys Ile Thr Lys Ala
50 55 60
Met Lys Met Val Ala Ala Ser Lys Leu Arg Ala Val Gln Gly Ala Pro
65 70 75 80
Ser Gln Thr Ile Asp Lys Ile Asn Ser Cys Tyr Pro Leu Phe Glu Gln
85 90 95
Asp Glu Tyr Gln Glu Leu Phe Arg Asn Lys Arg Gln Leu Glu Glu Ala
100 105 110
His Asp Ala Gln Arg Val Gln Glu Val Phe Ala Trp Thr Thr Thr Ala
115 120 125
Glu Tyr Glu Ala Leu Asn Phe Arg Arg Glu Ala Leu Thr Val Asp Pro
130 135 140
Ala Lys Ala Cys Gln Pro Leu Gly Ala Val Leu Cys Ser Leu Gly Phe
145 150 155 160
Ala Asn Thr Leu Pro Tyr Val His Gly Ser Gln Gly Cys Val Ala Tyr
165 170 175
Phe Arg Thr Tyr Phe Asn Arg His Phe Lys Glu Pro Ile Ala Cys Val
180 185 190
Ser Asp Ser Met Thr Glu Asp Ala Ala Val Phe Gly Gly Asn Asn Asn
195 200 205
Met Asn Leu Gly Leu Gln Asn Ala Ser Ala Leu Tyr Lys Pro Glu Ile
210 215 220
Ile Ala Val Ser Thr Thr Cys Met Ala Glu Val Ile Gly Asp Asp Leu
225 230 235 240
Gln Ala Phe Ile Ala Asn Ala Lys Lys Asp Gly Phe Val Asp Ser Ser
245 250 255
Ile Ala Val Pro His Ala His Thr Pro Ser Phe Ile Gly Ser His Val
260 265 270
Thr Gly Trp Asp Asn Met Phe Glu Gly Phe Ala Lys Thr Phe Thr Ala
275 280 285
Asp Tyr Gln Gly Gln Pro Gly Lys Leu Pro Lys Leu Asn Leu Val Thr
290 295 300
Gly Phe Glu Thr Tyr Leu Gly Asn Phe Arg Val Leu Lys Arg Met Met
305 310 315 320
Glu Gln Met Ala Val Pro Cys Ser Leu Leu Ser Asp Pro Ser Glu Val
325 330 335
Leu Asp Thr Pro Ala Asp Gly His Tyr Arg Met Tyr Ser Gly Gly Thr
340 345 350
Thr Gln Gln Glu Met Lys Glu Ala Pro Asp Ala Ile Asp Thr Leu Leu
355 360 365
Leu Gln Pro Trp Gln Leu Leu Lys Ser Lys Lys Val Val Gln Glu Met
370 375 380
Trp Asn Gln Pro Ala Thr Glu Val Ala Ile Pro Leu Gly Leu Ala Ala
385 390 395 400
Thr Asp Glu Leu Leu Met Thr Val Ser Gln Leu Ser Gly Lys Pro Ile
405 410 415
Ala Asp Ala Leu Thr Leu Glu Arg Gly Arg Leu Val Asp Met Met Leu
420 425 430
Asp Ser His Thr Trp Leu His Gly Lys Lys Phe Gly Leu Tyr Gly Asp
435 440 445
Pro Asp Phe Val Met Gly Leu Thr Arg Phe Leu Leu Glu Leu Gly Cys
450 455 460
Glu Pro Thr Val Ile Leu Ser His Asn Ala Asn Lys Arg Trp Gln Lys
465 470 475 480
Ala Met Asn Lys Met Leu Asp Ala Ser Pro Tyr Gly Arg Asp Ser Glu
485 490 495
Val Phe Ile Asn Cys Asp Leu Trp His Phe Arg Ser Leu Met Phe Thr
500 505 510
Arg Gln Pro Asp Phe Met Ile Gly Asn Ser Tyr Gly Lys Phe Ile Gln
515 520 525
Arg Asp Thr Leu Ala Lys Gly Lys Ala Phe Glu Val Pro Leu Ile Arg
530 535 540
Leu Gly Phe Pro Leu Phe Asp Arg His His Leu His Arg Gln Thr Thr
545 550 555 560
Trp Gly Tyr Glu Gly Ala Met Asn Ile Val Thr Thr Leu Val Asn Ala
565 570 575
Val Leu Glu Lys Leu Asp Ser Asp Thr Ser Gln Leu Gly Lys Thr Asp
580 585 590
Tyr Ser Phe Asp Leu Val Arg
595
<210> 58
<211> 4
<212> PRT
<213> Klebsiella oxytoca
<400> 58
Asp Leu Val Arg
1
<210> 59
<211> 60
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность мутантного полипептида MTP-FAv51
<400> 59
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ala Ala Ala Ala Ala Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Ala Ala Ala Ala Ala Ala Ala Ala Val
35 40 45
Val Arg Asn Arg Gly Gly Met Met Thr Asn Ala Thr
50 55 60
<210> 60
<211> 6
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 60
Val Arg Gly Cys Ala Tyr
1. 5
<210> 61
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 61
Arg Ala Gly Arg Arg Asn Tyr Tyr Thr Gly
1. 5 10
<210> 62
<211> 5
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 62
Ser Asn Arg Lys Ser
1. 5
<210> 63
<211> 5
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 63
Ala Ala Ala Ala Ala
1. 5
<210> 64
<211> 5
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 64
Gln Pro Gly Val Met
1. 5
<210> 65
<211> 5
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 65
Thr Val Arg Gly Cys
1. 5
<210> 66
<211> 5
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 66
Ala Tyr Ala Gly Ser
1. 5
<210> 67
<211> 5
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 67
Gly Ala Gly Ala Ala
1. 5
<210> 68
<211> 5
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 68
Lys Gly Val Val Phe
1. 5
<210> 69
<211> 5
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 69
Gly Pro Ile Lys Asp
1. 5
<210> 70
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 70
Thr Val Arg Gly Cys Ala Tyr Ala Gly Ser
1. 5 10
<210> 71
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 71
Thr Ala Arg Ala Cys Gly Tyr Gly Gly Ser
1. 5 10
<210> 72
<211> 4
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 72
Ala Tyr Ala Gly
1
<210> 73
<211> 4
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 73
Gly Ala Gly Gly
1
<210> 74
<211> 5
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 74
Met Ala His Ile Ser
1. 5
<210> 75
<211> 5
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 75
Ala Gly Ala Ala Ala
1. 5
<210> 76
<211> 5
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 76
His Gly Pro Val Gly
1. 5
<210> 77
<211> 5
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 77
Cys Gly Gln Tyr Ser
1. 5
<210> 78
<211> 5
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 78
Arg Ala Gly Arg Arg
1. 5
<210> 79
<211> 5
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 79
Asn Tyr Tyr Thr Gly
1. 5
<210> 80
<211> 5
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 80
Val Ser Gly Val Asp
1. 5
<210> 81
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 81
Arg Ala Gly Arg Arg Asn Tyr Phe Thr Gly
1. 5 10
<210> 82
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 82
Arg Ala Gly Arg Arg Asn Tyr Tyr Ala Gly
1. 5 10
<210> 83
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 83
Arg Ala Gly Arg Arg Asn Tyr Phe Ala Gly
1. 5 10
<210> 84
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 84
Arg Ala Gly Arg Arg Asn Tyr Tyr Ala Ala
1. 5 10
<210> 85
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 85
Arg Ala Gly Arg Ala Asn Tyr Tyr Thr Gly
1. 5 10
<210> 86
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 86
Arg Ala Gly Arg Arg His Tyr Tyr Thr Gly
1. 5 10
<210> 87
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 87
Arg Ala Gly Arg Arg Asn Gln Tyr Thr Gly
1. 5 10
<210> 88
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 88
Arg Ala Gly Arg Arg Asn Tyr Thr Thr Gly
1. 5 10
<210> 89
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 89
Arg Ala Gly Arg Arg Asn Tyr Tyr Val Gly
1. 5 10
<210> 90
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 90
Arg Ala Gly Arg Arg Asn Gln Thr Thr Gly
1. 5 10
<210> 91
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 91
Arg Ala Gly Arg Arg His Lys Gly Thr Gly
1. 5 10
<210> 92
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 92
Arg Ala Gly Arg Arg Asn Lys Tyr Thr Gly
1. 5 10
<210> 93
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 93
Arg Ala Gly Arg Arg Asn Lys Ala Thr Gly
1. 5 10
<210> 94
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 94
Arg Ala Gly Arg Arg Asn Tyr Ala Thr Gly
1. 5 10
<210> 95
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 95
Arg Ala Gly Arg Lys Asn Tyr Tyr Thr Gly
1. 5 10
<210> 96
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 96
Arg Ala Gly Arg Lys Asn Tyr Phe Thr Gly
1. 5 10
<210> 97
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 97
Arg Ala Gly Arg Lys Asn Tyr Tyr Ala Gly
1. 5 10
<210> 98
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 98
Arg Ala Gly Arg Lys Asn Tyr Phe Ala Gly
1. 5 10
<210> 99
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 99
Arg Ala Gly Arg Lys Asn Tyr Ala Ala Gly
1. 5 10
<210> 100
<211> 23
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 100
Tyr Tyr Thr Gly Val Ser Gly Val Asp Ser Phe Gly Thr Leu Asn Phe
1. 5 10 15
Thr Ser Asp Phe Gln Glu Arg
20
<210> 101
<211> 4
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 101
Arg Arg Asn Tyr
1
<210> 102
<211> 5
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 102
Arg Arg Asn Tyr Tyr
1. 5
<210> 103
<211> 4
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 103
Arg Arg Asn Gln
1
<210> 104
<211> 4
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 104
Arg Arg Asn Lys
1
<210> 105
<211> 5
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 105
Arg Arg Asn Gln Thr
1. 5
<210> 106
<211> 4
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 106
Arg Arg Phe Lys
1
<210> 107
<211> 5
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептид
<400> 107
Arg Arg Asn Lys Ala
1. 5
<210> 108
<211> 56
<212> DNA
<213> Искусственная последовательность
<220>
<223> Олигонуклеотидный праймер
<400> 108
ctaatgctac cggtgaacgt aacctggcac tgattcaaga agtactggaa gtgttc 56
<210> 109
<211> 57
<212> DNA
<213> Искусственная последовательность
<220>
<223> Олигонуклеотидный праймер
<400> 109
gttacgttca ccggtagcat tagtcatcat ccggctcctc cgctagataa aaatgtg 57
<210> 110
<211> 55
<212> DNA
<213> Искусственная последовательность
<220>
<223> Олигонуклеотидный праймер
<400> 110
gtttctggcg tcgactcttt cggcacgctg aacttcacct ctgacttcca ggaac 55
<210> 111
<211> 52
<212> DNA
<213> Искусственная последовательность
<220>
<223> Олигонуклеотидный праймер
<400> 111
cgaaagagtc gacgccagaa acgcccgtgt agtagttacg acgtcccgcg cg 52
<210> 112
<211> 26
<212> DNA
<213> Искусственная последовательность
<220>
<223> Олигонуклеотидный праймер
<400> 112
gaccaatgct accggtgaga ggaacc 26
<210> 113
<211> 29
<212> DNA
<213> Искусственная последовательность
<220>
<223> Олигонуклеотидный праймер
<400> 113
gttaagagtc ccgaaagagt cgacaccag 29
<210> 114
<211> 11
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность участка из 11 остатков из известной
неструктурированной линкерной области из целлобиогидролазы II Hypocrea
jecorina (Учетный №. AAG39980.1).
<400> 114
Ala Thr Pro Pro Pro Gly Ser Thr Thr Thr Arg
1. 5 10
<210> 115
<211> 9
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность из 9-остатков эпитопа HA
<400> 115
Pro Tyr Asp Val Pro Asp Tyr Ala
1. 5
<210> 116
<211> 30
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность линкера для слитого полипептида
NifD::линкер::NifK
<400> 116
Ala Thr Pro Pro Pro Gly Ser Thr Thr Thr Ala Tyr Pro Tyr Asp Val
1. 5 10 15
Pro Asp Tyr Ala Thr Pro Pro Pro Gly Ser Thr Thr Thr Ala
20 25 30
<210> 117
<211> 47
<212> DNA
<213> Искусственная последовательность
<220>
<223> Олигонуклеотидный праймер
<400> 117
gtcgtaacca atacacgggc gtttctggcg tcgactcttt cggcacg 47
<210> 118
<211> 41
<212> DNA
<213> Искусственная последовательность
<220>
<223> Олигонуклеотидный праймер
<400> 118
gcccgtgtat tggttacgac gtcccgcgcg agagtactgg c 41
<210> 119
<211> 7
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептидная последовательность
<400> 119
Ser Thr Gln Val Val Arg
1. 5
<210> 120
<211> 8
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептидная последовательность
<400> 120
Ser Thr Gln Val Val Arg
1. 5
<210> 121
<211> 603
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида с метаксином,
Кодированная конструкцией SN197. Эпитоп TwinStrep соответствует
Аминокислотам 1-31, mTurquoise соответствует аминокислотам 32-273,
а TEV представляет собой участок расщепления аминокислот 274-282 и
метаксиновую последовательность аминокислот
<400> 121
Ser His Pro Gln Phe Glu Lys Gly Gly Gly Ser Gly Gly Gly Ser Gly
1. 5 10 15
Gly Ser Ala Trp Ser His Pro Gln Phe Glu Lys Gly Gly
20 25 30
Ser Lys Gly Glu Glu Leu Phe Thr Gly Val Val Pro Ile Leu Val Glu
35 40 45
Leu Asp Gly Asp Val Asn Gly His Lys Phe Ser Val Ser Gly Glu Gly
50 55 60
Glu Gly Asp Ala Thr Tyr Gly Lys Leu Thr Leu Lys Phe Ile Cys Thr
65 70 75 80
Thr Gly Lys Leu Pro Val Pro Trp Pro Thr Leu Val Thr Thr Leu Ser
85 90 95
Trp Gly Val Gln Cys Phe Ala Arg Tyr Pro Asp His Met Lys Gln His
100 105 110
Asp Phe Phe Lys Ser Ala Met Pro Glu Gly Tyr Val Gln Glu Arg Thr
115 120 125
Ile Phe Phe Lys Asp Asp Gly Asn Tyr Lys Thr Arg Ala Glu Val Lys
130 135 140
Phe Glu Gly Asp Thr Leu Val Asn Arg Ile Glu Leu Lys Gly Ile Asp
145 150 155 160
Phe Lys Glu Asp Gly Asn Ile Leu Gly His Lys Leu Glu Tyr Asn Tyr
165 170 175
Phe Ser Asp Asn Val Tyr Ile Thr Ala Asp Lys Gln Lys Asn Gly Ile
180 185 190
Lys Ala Asn Phe Lys Ile Arg His Asn Ile Glu Asp Gly Gly Val Gln
195 200 205
Leu Ala Asp His Tyr Gln Gln Asn Thr Pro Ile Gly Asp Gly Pro Val
210 215 220
Leu Leu Pro Asp Asn His Tyr Leu Ser Thr Gln Ser Lys Leu Ser Lys
225 230 235 240
Asp Pro Asn Glu Lys Arg Asp His Met Val Leu Leu Glu Phe Val Thr
245 250 255
Ala Ala Gly Ile Thr Leu Gly Met Asp Glu Leu Tyr Lys Gly Gly Gly
260 265 270
Gly Glu Asn Leu Tyr Phe Gln Gly Gly Gly Met Glu Glu Ala Lys Glu
275 280 285
Arg Glu Lys Leu Thr Leu Val Thr Arg Lys Ser Ser Phe Gly Leu Pro
290 295 300
Thr Ser Cys Pro Asn Cys Leu Pro Val Tyr Leu Tyr Leu Lys Phe Ser
305 310 315 320
Lys Thr Pro Phe Asp Leu Ala Phe Asn Leu Ile Asn Pro Asp Phe Gly
325 330 335
Gln Ile Pro Tyr Val Glu Ser Gly Thr Tyr Val Ala Tyr Asn Asn Glu
340 345 350
Lys Gly Gly Val Ile Arg Ser Leu Ile Glu Asp Gly Phe Val Asp Leu
355 360 365
Asp Ser Gln Val His Gly Ile Pro Glu Trp Val Ser Thr Lys Ala Met
370 375 380
Val Asp Ser Trp Leu Ala Asp Ala Ile Leu Tyr Glu Leu Trp Val Gly
385 390 395 400
Ser Asp Gly Ser Ser Ala His Lys Ile Tyr Phe Ser Asp Leu Pro Trp
405 410 415
Pro Leu Gly Lys Leu Leu Tyr Leu Lys Gln Val His Val Ala Lys Gln
420 425 430
Ile Leu Asp Ile Thr Lys Asp Asn Ala Glu Arg Arg Glu Glu Glu Ile
435 440 445
Tyr Arg Asn Ala Asn Asp Ala Phe Ser Ala Leu Ser Thr Arg Leu Gly
450 455 460
Glu Gln Ala Tyr Leu Phe Asp Asn Arg Pro Thr Ser Leu Asp Ala Val
465 470 475 480
Phe Leu Gly His Ala Leu Phe Thr Leu Tyr Ala Leu Pro Glu Asn Ser
485 490 495
Val Leu Arg Asn Lys Leu Leu Glu His Asp Asn Leu Val Arg Tyr Thr
500 505 510
Glu Lys His Lys Leu Glu Leu Val Asp Ser Ser Ala Ser Ser Ser Ser
515 520 525
Gly Thr Gln Ser Gln Ser Asp Pro Ser Ser Val Pro Arg Arg Pro Ser
530 535 540
Gln Trp Ser Ser Lys Pro Lys Ser Lys Pro Lys Arg Glu Lys Thr Glu
545 550 555 560
Glu Glu Lys Lys Phe Arg Arg Arg Ala Lys Tyr Phe Leu Val Thr Gln
565 570 575
Leu Val Ala Val Leu Val Phe Leu Ser Leu Leu Gly Gly Ser Gly Asp
580 585 590
Ala Glu Val Glu Leu Asp Glu Asp Asp Tyr Glu
595 600
<210> 122
<211> 547
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-FA
51::NifH::HA, кодированная SN10. Аминокислоты 1-54 соответствуют
MTP-FA 51 с GG на ее C-конце, аминокислоты 55-536 представляли собой
аминокислоты NifD K. oxytoca(SEQ ID NO:18) с ее инициатором Met, и
амино
<400> 122
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly Met Met Thr Asn Ala Thr Gly Glu Arg Asn
50 55 60
Leu Ala Leu Ile Gln Glu Val Leu Glu Val Phe Pro Glu Thr Ala Arg
65 70 75 80
Lys Glu Arg Arg Lys His Met Met Val Ser Asp Pro Glu Met Glu Ser
85 90 95
Val Gly Lys Cys Ile Ile Ser Asn Arg Lys Ser Gln Pro Gly Val Met
100 105 110
Thr Val Arg Gly Cys Ala Tyr Ala Gly Ser Lys Gly Val Val Phe Gly
115 120 125
Pro Ile Lys Asp Met Ala His Ile Ser His Gly Pro Val Gly Cys Gly
130 135 140
Gln Tyr Ser Arg Ala Gly Arg Arg Asn Tyr Tyr Thr Gly Val Ser Gly
145 150 155 160
Val Asp Ser Phe Gly Thr Leu Asn Phe Thr Ser Asp Phe Gln Glu Arg
165 170 175
Asp Ile Val Phe Gly Gly Asp Lys Lys Leu Ser Lys Leu Ile Glu Glu
180 185 190
Met Glu Leu Leu Phe Pro Leu Thr Lys Gly Ile Thr Ile Gln Ser Glu
195 200 205
Cys Pro Val Gly Leu Ile Gly Asp Asp Ile Ser Ala Val Ala Asn Ala
210 215 220
Ser Ser Lys Ala Leu Asp Lys Pro Val Ile Pro Val Arg Cys Glu Gly
225 230 235 240
Phe Arg Gly Val Ser Gln Ser Leu Gly His His Ile Ala Asn Asp Val
245 250 255
Val Arg Asp Trp Ile Leu Asn Asn Arg Glu Gly Gln Pro Phe Glu Thr
260 265 270
Thr Pro Tyr Asp Val Ala Ile Ile Gly Asp Tyr Asn Ile Gly Gly Asp
275 280 285
Ala Trp Ala Ser Arg Ile Leu Leu Glu Glu Met Gly Leu Arg Val Val
290 295 300
Ala Gln Trp Ser Gly Asp Gly Thr Leu Val Glu Met Glu Asn Thr Pro
305 310 315 320
Phe Val Lys Leu Asn Leu Val His Cys Tyr Arg Ser Met Asn Tyr Ile
325 330 335
Ala Arg His Met Glu Glu Lys His Gln Ile Pro Trp Met Glu Tyr Asn
340 345 350
Phe Phe Gly Pro Thr Lys Ile Ala Glu Ser Leu Arg Lys Ile Ala Asp
355 360 365
Gln Phe Asp Asp Thr Ile Arg Ala Asn Ala Glu Ala Val Ile Ala Arg
370 375 380
Tyr Glu Gly Gln Met Ala Ala Ile Ile Ala Lys Tyr Arg Pro Arg Leu
385 390 395 400
Glu Gly Arg Lys Val Leu Leu Tyr Met Gly Gly Leu Arg Pro Arg His
405 410 415
Val Ile Gly Ala Tyr Glu Asp Leu Gly Met Glu Ile Ile Ala Ala Gly
420 425 430
Tyr Glu Phe Ala His Asn Asp Asp Tyr Asp Arg Thr Leu Pro Asp Leu
435 440 445
Lys Glu Gly Thr Leu Leu Phe Asp Asp Ala Ser Ser Tyr Glu Leu Glu
450 455 460
Ala Phe Val Lys Ala Leu Lys Pro Asp Leu Ile Gly Ser Gly Ile Lys
465 470 475 480
Glu Lys Tyr Ile Phe Gln Lys Met Gly Val Pro Phe Arg Gln Met His
485 490 495
Ser Trp Asp Tyr Ser Gly Pro Tyr His Gly Tyr Asp Gly Phe Ala Ile
500 505 510
Phe Ala Arg Asp Met Asp Met Thr Leu Asn Asn Pro Ala Trp Asn Glu
515 520 525
Leu Thr Ala Pro Trp Leu Lys Ser Gly Gly Tyr Pro Tyr Asp Val Pro
530 535 540
Asp Tyr Ala
545
<210> 123
<211> 331
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-FA
51::NifM::HA, кодированная SN30. Аминокислоты 1-54 соответствуют
MTP-FA 51 с GG на ее C-конце, аминокислоты 55-320 соответствуют NifM
K.оxytoca (SEQ ID NO:8) с ее инициатором Met, и амино
<400> 123
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly Met Asn Pro Trp Gln Arg Phe Ala Arg Gln
50 55 60
Arg Leu Ala Arg Ser Arg Trp Asn Arg Asp Pro Ala Ala Leu Asp Pro
65 70 75 80
Ala Asp Thr Pro Ala Phe Glu Gln Ala Trp Gln Arg Gln Cys His Met
85 90 95
Glu Gln Thr Ile Val Ala Arg Val Pro Glu Gly Asp Ile Pro Ala Ala
100 105 110
Leu Leu Glu Asn Ile Ala Ala Ser Leu Ala Ile Trp Leu Asp Glu Gly
115 120 125
Asp Phe Ala Pro Pro Glu Arg Ala Ala Ile Val Arg His His Ala Arg
130 135 140
Leu Glu Leu Ala Phe Ala Asp Ile Ala Arg Gln Ala Pro Gln Pro Asp
145 150 155 160
Leu Ser Thr Val Gln Ala Trp Tyr Leu Arg His Gln Thr Gln Phe Met
165 170 175
Arg Pro Glu Gln Arg Leu Thr Arg His Leu Leu Leu Thr Val Asp Asn
180 185 190
Asp Arg Glu Ala Val His Gln Arg Ile Leu Gly Leu Tyr Arg Gln Ile
195 200 205
Asn Ala Ser Arg Asp Ala Phe Ala Pro Leu Ala Gln Arg His Ser His
210 215 220
Cys Pro Ser Ala Leu Glu Glu Gly Arg Leu Gly Trp Ile Ser Arg Gly
225 230 235 240
Leu Leu Tyr Pro Gln Leu Glu Thr Ala Leu Phe Ser Leu Ala Glu Asn
245 250 255
Ala Leu Ser Leu Pro Ile Ala Ser Glu Leu Gly Trp His Leu Leu Trp
260 265 270
Cys Glu Ala Ile Arg Pro Ala Ala Pro Met Glu Pro Gln Gln Ala Leu
275 280 285
Glu Ser Ala Arg Asp Tyr Leu Trp Gln Gln Ser Gln Gln Arg His Gln
290 295 300
Arg Gln Trp Leu Glu Gln Met Ile Ser Arg Gln Pro Gly Leu Cys Gly
305 310 315 320
Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala
325 330
<210> 124
<211> 465
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-FA
51::NifS::HA, кодированная SN31. Аминокислоты 1-54 соответствуют
MTP-FA 51 с GG на ее C-конце, аминокислоты 55-454 соответствуют NifS
K.оxytoca (SEQ ID NO:19) с ее инициатором Met, соответственно
<400> 124
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly Met Lys Gln Val Tyr Leu Asp Asn Asn Ala
50 55 60
Thr Thr Arg Leu Asp Pro Met Val Leu Glu Ala Met Met Pro Phe Leu
65 70 75 80
Thr Asp Phe Tyr Gly Asn Pro Ser Ser Ile His Asp Phe Gly Ile Pro
85 90 95
Ala Gln Ala Ala Leu Glu Arg Ala His Gln Gln Ala Ala Ala Leu Leu
100 105 110
Gly Ala Glu Tyr Pro Ser Glu Ile Ile Phe Thr Ser Cys Ala Thr Glu
115 120 125
Ala Thr Ala Thr Ala Ile Ala Ser Ala Ile Ala Leu Leu Pro Glu Arg
130 135 140
Arg Glu Ile Ile Thr Ser Val Val Glu His Pro Ala Thr Leu Ala Ala
145 150 155 160
Cys Glu His Leu Glu Arg Gln Gly Tyr Arg Ile His Arg Ile Ala Val
165 170 175
Asp Ser Glu Gly Ala Leu Asp Met Ala Gln Phe Arg Ala Ala Leu Ser
180 185 190
Pro Arg Val Ala Leu Val Ser Val Met Trp Ala Asn Asn Glu Thr Gly
195 200 205
Val Leu Phe Pro Ile Gly Glu Met Ala Glu Leu Ala His Glu Gln Gly
210 215 220
Ala Leu Phe His Cys Asp Ala Val Gln Val Val Gly Lys Ile Pro Ile
225 230 235 240
Ala Val Gly Gln Thr Arg Ile Asp Met Leu Ser Cys Ser Ala His Lys
245 250 255
Phe His Gly Pro Lys Gly Val Gly Cys Leu Tyr Leu Arg Arg Gly Thr
260 265 270
Arg Phe Arg Pro Leu Leu Arg Gly Gly His Gln Glu Tyr Gly Arg Arg
275 280 285
Ala Gly Thr Glu Asn Ile Cys Gly Ile Val Gly Met Gly Ala Ala Cys
290 295 300
Glu Leu Ala Asn Ile His Leu Pro Gly Met Thr His Ile Gly Gln Leu
305 310 315 320
Arg Asn Arg Leu Glu His Arg Leu Leu Ala Ser Val Pro Ser Val Met
325 330 335
Val Met Gly Gly Gly Gln Pro Arg Val Pro Gly Thr Val Asn Leu Ala
340 345 350
Phe Glu Phe Ile Glu Gly Glu Ala Ile Leu Leu Leu Leu Asn Gln Ala
355 360 365
Gly Ile Ala Ala Ser Ser Gly Ser Ala Cys Thr Ser Gly Ser Leu Glu
370 375 380
Pro Ser His Val Met Arg Ala Met Asn Ile Pro Tyr Thr Ala Ala His
385 390 395 400
Gly Thr Ile Arg Phe Ser Leu Ser Arg Tyr Thr Arg Glu Lys Glu Ile
405 410 415
Asp Tyr Val Val Ala Thr Leu Pro Pro Ile Ile Asp Arg Leu Arg Ala
420 425 430
Leu Ser Pro Tyr Trp Gln Asn Gly Lys Pro Arg Pro Ala Asp Ala Val
435 440 445
Phe Thr Pro Val Tyr Gly Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr
450 455 460
Ala
465
<210> 125
<211> 339
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-FA
51::NifU::HA, кодированная SN32. Аминокислоты 1-54 соответствуют
MTP-FA 51 с GG на ее C-конце, аминокислоты 55-328 соответствуют
Люочнещсф ТшаГ (ЫУЙ ШВ ТЩЖ12) с ее инициатором Ьуеб и амино
<400> 125
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly Met Trp Asn Tyr Ser Glu Lys Val Lys Asp
50 55 60
His Phe Phe Asn Pro Arg Asn Ala Arg Val Val Asp Asn Ala Asn Ala
65 70 75 80
Val Gly Asp Val Gly Ser Leu Ser Cys Gly Asp Ala Leu Arg Leu Met
85 90 95
Leu Arg Val Asp Pro Gln Ser Glu Ile Ile Glu Glu Ala Gly Phe Gln
100 105 110
Thr Phe Gly Cys Gly Ser Ala Ile Ala Ser Ser Ser Ala Leu Thr Glu
115 120 125
Leu Ile Ile Gly His Thr Leu Ala Glu Ala Gly Gln Ile Thr Asn Gln
130 135 140
Gln Ile Ala Asp Tyr Leu Asp Gly Leu Pro Pro Glu Lys Met His Cys
145 150 155 160
Ser Val Met Gly Gln Glu Ala Leu Arg Ala Ala Ile Ala Asn Phe Arg
165 170 175
Gly Glu Ser Leu Glu Glu Glu His Asp Glu Gly Lys Leu Ile Cys Lys
180 185 190
Cys Phe Gly Val Asp Glu Gly His Ile Arg Arg Ala Val Gln Asn Asn
195 200 205
Gly Leu Thr Thr Leu Ala Glu Val Ile Asn Tyr Thr Lys Ala Gly Gly
210 215 220
Gly Cys Thr Ser Cys His Glu Lys Ile Glu Leu Ala Leu Ala Glu Ile
225 230 235 240
Leu Ala Gln Gln Pro Gln Thr Thr Pro Ala Val Ala Ser Gly Lys Asp
245 250 255
Pro His Trp Gln Ser Val Val Asp Thr Ile Ala Glu Leu Arg Pro His
260 265 270
Ile Gln Ala Asp Gly Gly Asp Met Ala Leu Leu Ser Val Thr Asn His
275 280 285
Gln Val Thr Val Ser Leu Ser Gly Ser Cys Ser Gly Cys Met Met Thr
290 295 300
Asp Met Thr Leu Ala Trp Leu Gln Gln Lys Leu Met Glu Arg Thr Gly
305 310 315 320
Cys Tyr Met Glu Val Val Ala Ala Gly Gly Tyr Pro Tyr Asp Val Pro
325 330 335
Asp Tyr Ala
<210> 126
<211> 522
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-FA
51::NifE::HA, кодированная SN38. Аминокислоты 1-54 соответствуют
MTP-FA 51 с GG на ее C-конце, аминокислоты 55-511 соответствуют
NifE K.оxytoca с ее инициатором Met согласно работы Temme et al.,
<400> 126
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly Met Lys Gly Asn Glu Ile Leu Ala Leu Leu
50 55 60
Asp Glu Pro Ala Cys Glu His Asn His Lys Gln Lys Ser Gly Cys Ser
65 70 75 80
Ala Pro Lys Pro Gly Ala Thr Ala Gly Gly Cys Ala Phe Asp Gly Ala
85 90 95
Gln Ile Thr Leu Leu Pro Ile Ala Asp Val Ala His Leu Val His Gly
100 105 110
Pro Ile Gly Cys Ala Gly Ser Ser Trp Asp Asn Arg Gly Ser Ala Ser
115 120 125
Ser Gly Pro Thr Leu Asn Arg Leu Gly Phe Thr Thr Asp Leu Asn Glu
130 135 140
Gln Asp Val Ile Met Gly Arg Gly Glu Arg Arg Leu Phe His Ala Val
145 150 155 160
Arg His Ile Val Thr Arg Tyr His Pro Ala Ala Val Phe Ile Tyr Asn
165 170 175
Thr Cys Val Pro Ala Met Glu Gly Asp Asp Leu Glu Ala Val Cys Gln
180 185 190
Ala Ala Gln Thr Ala Thr Gly Val Pro Val Ile Ala Ile Asp Ala Ala
195 200 205
Gly Phe Tyr Gly Ser Lys Asn Leu Gly Asn Arg Leu Ala Gly Asp Val
210 215 220
Met Val Lys Arg Val Ile Gly Gln Arg Glu Pro Ala Pro Trp Pro Glu
225 230 235 240
Ser Thr Leu Phe Ala Pro Glu Gln Arg His Asp Ile Gly Leu Ile Gly
245 250 255
Glu Phe Asn Ile Ala Gly Glu Phe Trp His Ile Gln Pro Leu Leu Asp
260 265 270
Glu Leu Gly Ile Arg Val Leu Gly Ser Leu Ser Gly Asp Gly Arg Phe
275 280 285
Ala Glu Ile Gln Thr Met His Arg Ala Gln Ala Asn Met Leu Val Cys
290 295 300
Ser Arg Ala Leu Ile Asn Val Ala Arg Ala Leu Glu Gln Arg Tyr Gly
305 310 315 320
Thr Pro Trp Phe Glu Gly Ser Phe Tyr Gly Ile Arg Ala Thr Ser Asp
325 330 335
Ala Leu Arg Gln Leu Ala Ala Leu Leu Gly Asp Asp Asp Leu Arg Gln
340 345 350
Arg Thr Glu Ala Leu Ile Ala Arg Glu Glu Gln Ala Ala Glu Leu Ala
355 360 365
Leu Gln Pro Trp Arg Glu Gln Leu Arg Gly Arg Lys Ala Leu Leu Tyr
370 375 380
Thr Gly Gly Val Lys Ser Trp Ser Val Val Ser Ala Leu Gln Asp Leu
385 390 395 400
Gly Met Thr Val Val Ala Thr Gly Thr Arg Lys Ser Thr Glu Glu Asp
405 410 415
Lys Gln Arg Ile Arg Glu Leu Met Gly Glu Glu Ala Val Met Leu Glu
420 425 430
Glu Gly Asn Ala Arg Thr Leu Leu Asp Val Val Tyr Arg Tyr Gln Ala
435 440 445
Asp Leu Met Ile Ala Gly Gly Arg Asn Met Tyr Thr Ala Tyr Lys Ala
450 455 460
Arg Leu Pro Phe Leu Asp Ile Asn Gln Glu Arg Glu His Ala Phe Ala
465 470 475 480
Gly Tyr Gln Gly Ile Val Thr Leu Ala Arg Gln Leu Cys Gln Thr Ile
485 490 495
Asn Ser Pro Ile Trp Pro Gln Thr His Ser Arg Ala Pro Trp Arg Gly
500 505 510
Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala
515 520
<210> 127
<211> 526
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-FA
51::NifN::HA, кодированная SN39. Аминокислоты 1-54 соответствуют
MTP-FA 51 с GG на ее C-конце, аминокислоты 55-515 соответствуют
ТшаТ Люочнещсф (ЫУЙ ШВ ТЩЖ9) с ее инициатором Ьуеб и амино
<400> 127
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly Met Ala Asp Ile Phe Arg Thr Asp Lys Pro
50 55 60
Leu Ala Val Ser Pro Ile Lys Thr Gly Gln Pro Leu Gly Ala Ile Leu
65 70 75 80
Ala Ser Leu Gly Ile Glu His Ser Ile Pro Leu Val His Gly Ala Gln
85 90 95
Gly Cys Ser Ala Phe Ala Lys Val Phe Phe Ile Gln His Phe His Asp
100 105 110
Pro Val Pro Leu Gln Ser Thr Ala Met Asp Pro Thr Ser Thr Ile Met
115 120 125
Gly Ala Asp Gly Asn Ile Phe Thr Ala Leu Asp Thr Leu Cys Gln Arg
130 135 140
Asn Asn Pro Gln Ala Ile Val Leu Leu Ser Thr Gly Leu Ser Glu Ala
145 150 155 160
Gln Gly Ser Asp Ile Ser Arg Val Val Arg Gln Phe Arg Glu Glu Tyr
165 170 175
Pro Arg His Lys Gly Val Ala Ile Leu Thr Val Asn Thr Pro Asp Phe
180 185 190
Tyr Gly Ser Met Glu Asn Gly Phe Ser Ala Val Leu Glu Ser Val Ile
195 200 205
Glu Gln Trp Val Pro Pro Ala Pro Arg Pro Ala Gln Arg Asn Arg Arg
210 215 220
Val Asn Leu Leu Val Ser His Leu Cys Ser Pro Gly Asp Ile Glu Trp
225 230 235 240
Leu Arg Arg Cys Val Glu Ala Phe Gly Leu Gln Pro Ile Ile Leu Pro
245 250 255
Asp Leu Ala Gln Ser Met Asp Gly His Leu Ala Gln Gly Asp Phe Ser
260 265 270
Pro Leu Thr Gln Gly Gly Thr Pro Leu Arg Gln Ile Glu Gln Met Gly
275 280 285
Gln Ser Leu Cys Ser Phe Ala Ile Gly Val Ser Leu His Arg Ala Ser
290 295 300
Ser Leu Leu Ala Pro Arg Cys Arg Gly Glu Val Ile Ala Leu Pro His
305 310 315 320
Leu Met Thr Leu Glu Arg Cys Asp Ala Phe Ile His Gln Leu Ala Lys
325 330 335
Ile Ser Gly Arg Ala Val Pro Glu Trp Leu Glu Arg Gln Arg Gly Gln
340 345 350
Leu Gln Asp Ala Met Ile Asp Cys His Met Trp Leu Gln Gly Gln Arg
355 360 365
Met Ala Ile Ala Ala Glu Gly Asp Leu Leu Ala Ala Trp Cys Asp Phe
370 375 380
Ala Asn Ser Gln Gly Met Gln Pro Gly Pro Leu Val Ala Pro Thr Gly
385 390 395 400
His Pro Ser Leu Arg Gln Leu Pro Val Glu Arg Val Val Pro Gly Asp
405 410 415
Leu Glu Asp Leu Gln Thr Leu Leu Cys Ala His Pro Ala Asp Leu Leu
420 425 430
Val Ala Asn Ser His Ala Arg Asp Leu Ala Glu Gln Phe Ala Leu Pro
435 440 445
Leu Val Arg Ala Gly Phe Pro Leu Phe Asp Lys Leu Gly Glu Phe Arg
450 455 460
Arg Val Arg Gln Gly Tyr Ser Gly Met Arg Asp Thr Leu Phe Glu Leu
465 470 475 480
Ala Asn Leu Ile Arg Glu Arg His His His Leu Ala His Tyr Arg Ser
485 490 495
Pro Leu Arg Gln Asn Pro Glu Ser Ser Leu Ser Thr Gly Gly Ala Tyr
500 505 510
Ala Ala Asp Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala
515 520 525
<210> 128
<211> 365
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-CoxIV-
TwinStrep::NifH::HA, кодированная SN42. Аминокислоты 1-61
соответствуют MTP-CoxIV-TwinStrep с GG ее C-конце, аминокислоты
62-354 представляли собой аминокислоты NifH K. Oxytoca (SEQ ID NO:1)с
<400> 128
Met Leu Ser Leu Arg Gln Ser Ile Arg Phe Phe Lys Pro Ala Thr Arg
1. 5 10 15
Thr Leu Cys Ser Ser Arg Tyr Leu Leu Gln Gln Lys Pro Ser Ala Trp
20 25 30
Ser His Pro Gln Phe Glu Lys Gly Gly Gly Ser Gly Gly Gly Ser Gly
35 40 45
Gly Ser Ala Trp Ser His Pro Gln Phe Glu Lys Gly Gly Met Thr Met
50 55 60
Arg Gln Cys Ala Ile Tyr Gly Lys Gly Gly Ile Gly Lys Ser Thr Thr
65 70 75 80
Thr Gln Asn Leu Val Ala Ala Leu Ala Glu Met Gly Lys Lys Val Met
85 90 95
Ile Val Gly Cys Asp Pro Lys Ala Asp Ser Thr Arg Leu Ile Leu His
100 105 110
Ala Lys Ala Gln Asn Thr Ile Met Glu Met Ala Ala Glu Val Gly Ser
115 120 125
Val Glu Asp Leu Glu Leu Glu Asp Val Leu Gln Ile Gly Tyr Gly Asp
130 135 140
Val Arg Cys Ala Glu Ser Gly Gly Pro Glu Pro Gly Val Gly Cys Ala
145 150 155 160
Gly Arg Gly Val Ile Thr Ala Ile Asn Phe Leu Glu Glu Glu Gly Ala
165 170 175
Tyr Glu Asp Asp Leu Asp Phe Val Phe Tyr Asp Val Leu Gly Asp Val
180 185 190
Val Cys Gly Gly Phe Ala Met Pro Ile Arg Glu Asn Lys Ala Gln Glu
195 200 205
Ile Tyr Ile Val Cys Ser Gly Glu Met Met Ala Met Tyr Ala Ala Asn
210 215 220
Asn Ile Ser Lys Gly Ile Val Lys Tyr Ala Lys Ser Gly Lys Val Arg
225 230 235 240
Leu Gly Gly Leu Ile Cys Asn Ser Arg Gln Thr Asp Arg Glu Asp Glu
245 250 255
Leu Ile Ile Ala Leu Ala Glu Lys Leu Gly Thr Gln Met Ile His Phe
260 265 270
Val Pro Arg Asp Asn Ile Val Gln Arg Ala Glu Ile Arg Arg Met Thr
275 280 285
Val Ile Glu Tyr Asp Pro Ala Cys Lys Gln Ala Asn Glu Tyr Arg Thr
290 295 300
Leu Ala Gln Lys Ile Val Asn Asn Thr Met Lys Val Val Pro Thr Pro
305 310 315 320
Cys Thr Met Asp Glu Leu Glu Ser Leu Leu Met Glu Phe Gly Ile Met
325 330 335
Glu Glu Glu Asp Thr Ser Ile Ile Gly Lys Thr Ala Ala Glu Glu Asn
340 345 350
Ala Ala Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala
355 360 365
<210> 129
<211> 590
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-Su9::NifK,
кодированная SN46. Аминокислоты 1-70 соответствуют MTP-Su9 с
GG на ее C-конце, аминокислоты 71-590 соответствуют NifK K.oxytoca
(SEQ ID NO:3) с ее инициатором Met
<400> 129
Met Ala Ser Thr Arg Val Leu Ala Ser Arg Leu Ala Ser Gln Met Ala
1. 5 10 15
Ala Ser Ala Lys Val Ala Arg Pro Ala Val Arg Val Ala Gln Val Ser
20 25 30
Lys Arg Thr Ile Gln Thr Gly Ser Pro Leu Gln Thr Leu Lys Arg Thr
35 40 45
Gln Met Thr Ser Ile Val Asn Ala Thr Thr Arg Gln Ala Phe Gln Lys
50 55 60
Arg Ala Tyr Ser Gly Gly Met Ser Gln Thr Ile Asp Lys Ile Asn Ser
65 70 75 80
Cys Tyr Pro Leu Phe Glu Gln Asp Glu Tyr Gln Glu Leu Phe Arg Asn
85 90 95
Lys Arg Gln Leu Glu Glu Ala His Asp Ala Gln Arg Val Gln Glu Val
100 105 110
Phe Ala Trp Thr Thr Thr Ala Glu Tyr Glu Ala Leu Asn Phe Gln Arg
115 120 125
Glu Ala Leu Thr Val Asp Pro Ala Lys Ala Cys Gln Pro Leu Gly Ala
130 135 140
Val Leu Cys Ser Leu Gly Phe Ala Asn Thr Leu Pro Tyr Val His Gly
145 150 155 160
Ser Gln Gly Cys Val Ala Tyr Phe Arg Thr Tyr Phe Asn Arg His Phe
165 170 175
Lys Glu Pro Ile Ala Cys Val Ser Asp Ser Met Thr Glu Asp Ala Ala
180 185 190
Val Phe Gly Gly Asn Asn Asn Met Asn Leu Gly Leu Gln Asn Ala Ser
195 200 205
Ala Leu Tyr Lys Pro Glu Ile Ile Ala Val Ser Thr Thr Cys Met Ala
210 215 220
Glu Val Ile Gly Asp Asp Leu Gln Ala Phe Ile Ala Asn Ala Lys Lys
225 230 235 240
Asp Gly Phe Val Asp Ser Ser Ile Ala Val Pro His Ala His Thr Pro
245 250 255
Ser Phe Ile Gly Ser His Val Thr Gly Trp Asp Asn Met Phe Glu Gly
260 265 270
Phe Ala Lys Thr Phe Thr Ala Asp Tyr Gln Gly Gln Pro Gly Lys Leu
275 280 285
Pro Lys Leu Asn Leu Val Thr Gly Phe Glu Thr Tyr Leu Gly Asn Phe
290 295 300
Arg Val Leu Lys Arg Met Met Glu Gln Met Ala Val Pro Cys Ser Leu
305 310 315 320
Leu Ser Asp Pro Ser Glu Val Leu Asp Thr Pro Ala Asp Gly His Tyr
325 330 335
Arg Met Tyr Ser Gly Gly Thr Thr Gln Gln Glu Met Lys Glu Ala Pro
340 345 350
Asp Ala Ile Asp Thr Leu Leu Leu Gln Pro Trp Gln Leu Leu Lys Ser
355 360 365
Lys Lys Val Val Gln Glu Met Trp Asn Gln Pro Ala Thr Glu Val Ala
370 375 380
Ile Pro Leu Gly Leu Ala Ala Thr Asp Glu Leu Leu Met Thr Val Ser
385 390 395 400
Gln Leu Ser Gly Lys Pro Ile Ala Asp Ala Leu Thr Leu Glu Arg Gly
405 410 415
Arg Leu Val Asp Met Met Leu Asp Ser His Thr Trp Leu His Gly Lys
420 425 430
Lys Phe Gly Leu Tyr Gly Asp Pro Asp Phe Val Met Gly Leu Thr Arg
435 440 445
Phe Leu Leu Glu Leu Gly Cys Glu Pro Thr Val Ile Leu Ser His Asn
450 455 460
Ala Asn Lys Arg Trp Gln Lys Ala Met Asn Lys Met Leu Asp Ala Ser
465 470 475 480
Pro Tyr Gly Arg Asp Ser Glu Val Phe Ile Asn Cys Asp Leu Trp His
485 490 495
Phe Arg Ser Leu Met Phe Thr Arg Gln Pro Asp Phe Met Ile Gly Asn
500 505 510
Ser Tyr Gly Lys Phe Ile Gln Arg Asp Thr Leu Ala Lys Gly Lys Ala
515 520 525
Phe Glu Val Pro Leu Ile Arg Leu Gly Phe Pro Leu Phe Asp Arg His
530 535 540
His Leu His Arg Gln Thr Thr Trp Gly Tyr Glu Gly Ala Met Asn Ile
545 550 555 560
Val Thr Thr Leu Val Asn Ala Val Leu Glu Lys Leu Asp Ser Asp Thr
565 570 575
Ser Gln Leu Gly Lys Thr Asp Tyr Ser Phe Asp Leu Val Arg
580 585 590
<210> 130
<211> 426
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-
L29::NifV::HA, кодированная SN51. Аминокислоты 1-34 соответствуют
MTP-L29 с GG на ее C-конце, аминокислоты 35-415 соответствуют NifV
K.oxytoca(SEQ ID NO:13) с ее инициатором Met, и аминоаминокислотам 416-
<400> 130
Met Phe Leu Thr Arg Phe Val Gly Arg Arg Phe Leu Ala Ala Ala Ser
1. 5 10 15
Ala Arg Ser Glu Ser Thr Thr Ala Ala Ala Ala Ala Ser Thr Ile Arg
20 25 30
Gly Gly Met Glu Arg Val Leu Ile Asn Asp Thr Thr Leu Arg Asp Gly
35 40 45
Glu Gln Ser Pro Gly Val Ala Phe Arg Thr Ser Glu Lys Val Ala Ile
50 55 60
Ala Glu Ala Leu Tyr Ala Ala Gly Ile Thr Ala Met Glu Val Gly Thr
65 70 75 80
Pro Ala Met Gly Asp Glu Glu Ile Ala Arg Ile Gln Leu Val Arg Arg
85 90 95
Gln Leu Pro Asp Ala Thr Leu Met Thr Trp Cys Arg Met Asn Ala Leu
100 105 110
Glu Ile Arg Gln Ser Ala Asp Leu Gly Ile Asp Trp Val Asp Ile Ser
115 120 125
Ile Pro Ala Ser Asp Lys Leu Arg Gln Tyr Lys Leu Arg Glu Pro Leu
130 135 140
Ala Val Leu Leu Glu Arg Leu Ala Met Phe Ile His Leu Ala His Thr
145 150 155 160
Leu Gly Leu Lys Val Cys Ile Gly Cys Glu Asp Ala Ser Arg Ala Ser
165 170 175
Gly Gln Thr Leu Arg Ala Ile Ala Glu Val Ala Gln Asn Ala Pro Ala
180 185 190
Ala Arg Leu Arg Tyr Ala Asp Thr Val Gly Leu Leu Asp Pro Phe Thr
195 200 205
Thr Ala Ala Gln Ile Ser Ala Leu Arg Asp Val Trp Ser Gly Glu Ile
210 215 220
Glu Met His Ala His Asn Asp Leu Gly Met Ala Thr Ala Asn Thr Leu
225 230 235 240
Ala Ala Val Ser Ala Gly Ala Thr Ser Val Asn Thr Thr Val Leu Gly
245 250 255
Leu Gly Glu Arg Ala Gly Asn Ala Ala Ala Trp Lys Pro Ser Ala Leu
260 265 270
Gly Leu Glu Arg Cys Leu Gly Val Glu Thr Gly Val His Phe Ser Ala
275 280 285
Leu Pro Ala Leu Cys Gln Arg Val Ala Glu Ala Ala Gln Arg Ala Ile
290 295 300
Asp Pro Gln Gln Pro Leu Val Gly Glu Leu Val Phe Thr His Glu Ser
305 310 315 320
Gly Val His Val Ala Ala Leu Leu Arg Asp Ser Glu Ser Tyr Gln Ser
325 330 335
Ile Ala Pro Ser Leu Met Gly Arg Ser Tyr Arg Leu Val Leu Gly Lys
340 345 350
His Ser Gly Arg Gln Ala Val Asn Gly Val Phe Asp Gln Met Gly Tyr
355 360 365
His Leu Asn Ala Ala Gln Ile Asn Gln Leu Leu Pro Ala Ile Arg Arg
370 375 380
Phe Ala Glu Asn Trp Lys Arg Ser Pro Lys Asp Tyr Glu Leu Val Ala
385 390 395 400
Ile Tyr Asp Glu Leu Cys Gly Glu Ser Ala Leu Arg Ala Arg Gly Gly
405 410 415
Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala
420 425
<210> 131
<211> 1085
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-FA
51::NifH::линкер(HA)::NifK, кодированная SN68. Аминокислоты 1-54
соответствуют MTP-FA 51 с GG на ее C-конце, аминокислоты 55-536
соответствуют аминокислотам NifD K.oxytoca дикого типа (SEQ ID NO:18)
<400> 131
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly Met Thr Asn Ala Thr Gly Glu Arg Asn Leu
50 55 60
Ala Leu Ile Gln Glu Val Leu Glu Val Phe Pro Glu Thr Ala Arg Lys
65 70 75 80
Glu Arg Arg Lys His Met Met Val Ser Asp Pro Glu Met Glu Ser Val
85 90 95
Gly Lys Cys Ile Ile Ser Asn Arg Lys Ser Gln Pro Gly Val Met Thr
100 105 110
Val Arg Gly Cys Ala Tyr Ala Gly Ser Lys Gly Val Val Phe Gly Pro
115 120 125
Ile Lys Asp Met Ala His Ile Ser His Gly Pro Val Gly Cys Gly Gln
130 135 140
Tyr Ser Arg Ala Gly Arg Arg Asn Tyr Tyr Thr Gly Val Ser Gly Val
145 150 155 160
Asp Ser Phe Gly Thr Leu Asn Phe Thr Ser Asp Phe Gln Glu Arg Asp
165 170 175
Ile Val Phe Gly Gly Asp Lys Lys Leu Ser Lys Leu Ile Glu Glu Met
180 185 190
Glu Leu Leu Phe Pro Leu Thr Lys Gly Ile Thr Ile Gln Ser Glu Cys
195 200 205
Pro Val Gly Leu Ile Gly Asp Asp Ile Ser Ala Val Ala Asn Ala Ser
210 215 220
Ser Lys Ala Leu Asp Lys Pro Val Ile Pro Val Arg Cys Glu Gly Phe
225 230 235 240
Arg Gly Val Ser Gln Ser Leu Gly His His Ile Ala Asn Asp Val Val
245 250 255
Arg Asp Trp Ile Leu Asn Asn Arg Glu Gly Gln Pro Phe Glu Thr Thr
260 265 270
Pro Tyr Asp Val Ala Ile Ile Gly Asp Tyr Asn Ile Gly Gly Asp Ala
275 280 285
Trp Ala Ser Arg Ile Leu Leu Glu Glu Met Gly Leu Arg Val Val Ala
290 295 300
Gln Trp Ser Gly Asp Gly Thr Leu Val Glu Met Glu Asn Thr Pro Phe
305 310 315 320
Val Lys Leu Asn Leu Val His Cys Tyr Arg Ser Met Asn Tyr Ile Ala
325 330 335
Arg His Met Glu Glu Lys His Gln Ile Pro Trp Met Glu Tyr Asn Phe
340 345 350
Phe Gly Pro Thr Lys Ile Ala Glu Ser Leu Arg Lys Ile Ala Asp Gln
355 360 365
Phe Asp Asp Thr Ile Arg Ala Asn Ala Glu Ala Val Ile Ala Arg Tyr
370 375 380
Glu Gly Gln Met Ala Ala Ile Ile Ala Lys Tyr Arg Pro Arg Leu Glu
385 390 395 400
Gly Arg Lys Val Leu Leu Tyr Met Gly Gly Leu Arg Pro Arg His Val
405 410 415
Ile Gly Ala Tyr Glu Asp Leu Gly Met Glu Ile Ile Ala Ala Gly Tyr
420 425 430
Glu Phe Ala His Asn Asp Asp Tyr Asp Arg Thr Leu Pro Asp Leu Lys
435 440 445
Glu Gly Thr Leu Leu Phe Asp Asp Ala Ser Ser Tyr Glu Leu Glu Ala
450 455 460
Phe Val Lys Ala Leu Lys Pro Asp Leu Ile Gly Ser Gly Ile Lys Glu
465 470 475 480
Lys Tyr Ile Phe Gln Lys Met Gly Val Pro Phe Arg Gln Met His Ser
485 490 495
Trp Asp Tyr Ser Gly Pro Tyr His Gly Tyr Asp Gly Phe Ala Ile Phe
500 505 510
Ala Arg Asp Met Asp Met Thr Leu Asn Asn Pro Ala Trp Asn Glu Leu
515 520 525
Thr Ala Pro Trp Leu Lys Ser Ala Ala Thr Pro Pro Pro Gly Ser Thr
530 535 540
Thr Thr Ala Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Thr Pro Pro Pro
545 550 555 560
Gly Ser Thr Thr Thr Ala Ser Gln Thr Ile Asp Lys Ile Asn Ser Cys
565 570 575
Tyr Pro Leu Phe Glu Gln Asp Glu Tyr Gln Glu Leu Phe Arg Asn Lys
580 585 590
Arg Gln Leu Glu Glu Ala His Asp Ala Gln Arg Val Gln Glu Val Phe
595 600 605
Ala Trp Thr Thr Thr Ala Glu Tyr Glu Ala Leu Asn Phe Gln Arg Glu
610 615 620
Ala Leu Thr Val Asp Pro Ala Lys Ala Cys Gln Pro Leu Gly Ala Val
625 630 635 640
Leu Cys Ser Leu Gly Phe Ala Asn Thr Leu Pro Tyr Val His Gly Ser
645 650 655
Gln Gly Cys Val Ala Tyr Phe Arg Thr Tyr Phe Asn Arg His Phe Lys
660 665 670
Glu Pro Ile Ala Cys Val Ser Asp Ser Met Thr Glu Asp Ala Ala Val
675 680 685
Phe Gly Gly Asn Asn Asn Met Asn Leu Gly Leu Gln Asn Ala Ser Ala
690 695 700
Leu Tyr Lys Pro Glu Ile Ile Ala Val Ser Thr Thr Cys Met Ala Glu
705 710 715 720
Val Ile Gly Asp Asp Leu Gln Ala Phe Ile Ala Asn Ala Lys Lys Asp
725 730 735
Gly Phe Val Asp Ser Ser Ile Ala Val Pro His Ala His Thr Pro Ser
740 745 750
Phe Ile Gly Ser His Val Thr Gly Trp Asp Asn Met Phe Glu Gly Phe
755 760 765
Ala Lys Thr Phe Thr Ala Asp Tyr Gln Gly Gln Pro Gly Lys Leu Pro
770 775 780
Lys Leu Asn Leu Val Thr Gly Phe Glu Thr Tyr Leu Gly Asn Phe Arg
785 790 795 800
Val Leu Lys Arg Met Met Glu Gln Met Ala Val Pro Cys Ser Leu Leu
805 810 815
Ser Asp Pro Ser Glu Val Leu Asp Thr Pro Ala Asp Gly His Tyr Arg
820 825 830
Met Tyr Ser Gly Gly Thr Thr Gln Gln Glu Met Lys Glu Ala Pro Asp
835 840 845
Ala Ile Asp Thr Leu Leu Leu Gln Pro Trp Gln Leu Leu Lys Ser Lys
850 855 860
Lys Val Val Gln Glu Met Trp Asn Gln Pro Ala Thr Glu Val Ala Ile
865 870 875 880
Pro Leu Gly Leu Ala Ala Thr Asp Glu Leu Leu Met Thr Val Ser Gln
885 890 895
Leu Ser Gly Lys Pro Ile Ala Asp Ala Leu Thr Leu Glu Arg Gly Arg
900 905 910
Leu Val Asp Met Met Leu Asp Ser His Thr Trp Leu His Gly Lys Lys
915 920 925
Phe Gly Leu Tyr Gly Asp Pro Asp Phe Val Met Gly Leu Thr Arg Phe
930 935 940
Leu Leu Glu Leu Gly Cys Glu Pro Thr Val Ile Leu Ser His Asn Ala
945 950 955 960
Asn Lys Arg Trp Gln Lys Ala Met Asn Lys Met Leu Asp Ala Ser Pro
965 970 975
Tyr Gly Arg Asp Ser Glu Val Phe Ile Asn Cys Asp Leu Trp His Phe
980 985 990
Arg Ser Leu Met Phe Thr Arg Gln Pro Asp Phe Met Ile Gly Asn Ser
995 1000 1005
Tyr Gly Lys Phe Ile Gln Arg Asp Thr Leu Ala Lys Gly Lys Ala
1010 1015 1020
Phe Glu Val Pro Leu Ile Arg Leu Gly Phe Pro Leu Phe Asp Arg
1025 1030 1035
His His Leu His Arg Gln Thr Thr Trp Gly Tyr Glu Gly Ala Met
1040 1045 1050
Asn Ile Val Thr Thr Leu Val Asn Ala Val Leu Glu Lys Leu Asp
1055 1060 1065
Ser Asp Thr Ser Gln Leu Gly Lys Thr Asp Tyr Ser Phe Asp Leu
1070 1075 1080
Val Arg
1085
<210> 132
<211> 557
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-FA
51::HA::NifD::HA, кодированная SN75. Аминокислоты 1-53 соответствуют
MTP-FA 51 с GG на ее C-конце, аминокислоты 54-64 соответствуют
первому эпитопу HA, аминокислоты 65-546 соответствуют
<400> 132
Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro Ser
1. 5 10 15
Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser Asp
20 25 30
Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val Val
35 40 45
Arg Asn Arg Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Gly Gly
50 55 60
Met Met Thr Asn Ala Thr Gly Glu Arg Asn Leu Ala Leu Ile Gln Glu
65 70 75 80
Val Leu Glu Val Phe Pro Glu Thr Ala Arg Lys Glu Arg Arg Lys His
85 90 95
Met Met Val Ser Asp Pro Glu Met Glu Ser Val Gly Lys Cys Ile Ile
100 105 110
Ser Asn Arg Lys Ser Gln Pro Gly Val Met Thr Val Arg Gly Cys Ala
115 120 125
Tyr Ala Gly Ser Lys Gly Val Val Phe Gly Pro Ile Lys Asp Met Ala
130 135 140
His Ile Ser His Gly Pro Val Gly Cys Gly Gln Tyr Ser Arg Ala Gly
145 150 155 160
Arg Arg Asn Tyr Tyr Thr Gly Val Ser Gly Val Asp Ser Phe Gly Thr
165 170 175
Leu Asn Phe Thr Ser Asp Phe Gln Glu Arg Asp Ile Val Phe Gly Gly
180 185 190
Asp Lys Lys Leu Ser Lys Leu Ile Glu Glu Met Glu Leu Leu Phe Pro
195 200 205
Leu Thr Lys Gly Ile Thr Ile Gln Ser Glu Cys Pro Val Gly Leu Ile
210 215 220
Gly Asp Asp Ile Ser Ala Val Ala Asn Ala Ser Ser Lys Ala Leu Asp
225 230 235 240
Lys Pro Val Ile Pro Val Arg Cys Glu Gly Phe Arg Gly Val Ser Gln
245 250 255
Ser Leu Gly His His Ile Ala Asn Asp Val Val Arg Asp Trp Ile Leu
260 265 270
Asn Asn Arg Glu Gly Gln Pro Phe Glu Thr Thr Pro Tyr Asp Val Ala
275 280 285
Ile Ile Gly Asp Tyr Asn Ile Gly Gly Asp Ala Trp Ala Ser Arg Ile
290 295 300
Leu Leu Glu Glu Met Gly Leu Arg Val Val Ala Gln Trp Ser Gly Asp
305 310 315 320
Gly Thr Leu Val Glu Met Glu Asn Thr Pro Phe Val Lys Leu Asn Leu
325 330 335
Val His Cys Tyr Arg Ser Met Asn Tyr Ile Ala Arg His Met Glu Glu
340 345 350
Lys His Gln Ile Pro Trp Met Glu Tyr Asn Phe Phe Gly Pro Thr Lys
355 360 365
Ile Ala Glu Ser Leu Arg Lys Ile Ala Asp Gln Phe Asp Asp Thr Ile
370 375 380
Arg Ala Asn Ala Glu Ala Val Ile Ala Arg Tyr Glu Gly Gln Met Ala
385 390 395 400
Ala Ile Ile Ala Lys Tyr Arg Pro Arg Leu Glu Gly Arg Lys Val Leu
405 410 415
Leu Tyr Met Gly Gly Leu Arg Pro Arg His Val Ile Gly Ala Tyr Glu
420 425 430
Asp Leu Gly Met Glu Ile Ile Ala Ala Gly Tyr Glu Phe Ala His Asn
435 440 445
Asp Asp Tyr Asp Arg Thr Leu Pro Asp Leu Lys Glu Gly Thr Leu Leu
450 455 460
Phe Asp Asp Ala Ser Ser Tyr Glu Leu Glu Ala Phe Val Lys Ala Leu
465 470 475 480
Lys Pro Asp Leu Ile Gly Ser Gly Ile Lys Glu Lys Tyr Ile Phe Gln
485 490 495
Lys Met Gly Val Pro Phe Arg Gln Met His Ser Trp Asp Tyr Ser Gly
500 505 510
Pro Tyr His Gly Tyr Asp Gly Phe Ala Ile Phe Ala Arg Asp Met Asp
515 520 525
Met Thr Leu Asn Asn Pro Ala Trp Asn Glu Leu Thr Ala Pro Trp Leu
530 535 540
Lys Ser Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala
545 550 555
<210> 133
<211> 547
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-FA
51::NifH::HA, кодированная SN99. Аминокислоты 1-54 соответствуют
MTP-FA 51 с GG на ее C-конце, аминокислоты 55-536 соответствуют
NifD K.oxytoca, содержащие мутации по типу замены аланина в
<400> 133
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly Met Met Thr Asn Ala Thr Gly Glu Arg Asn
50 55 60
Leu Ala Leu Ile Gln Glu Val Leu Glu Val Phe Pro Glu Thr Ala Arg
65 70 75 80
Lys Glu Arg Arg Lys His Met Met Val Ser Asp Pro Glu Met Glu Ser
85 90 95
Val Gly Lys Cys Ile Ile Ser Asn Arg Lys Ser Gln Pro Gly Val Met
100 105 110
Thr Val Arg Gly Cys Ala Tyr Ala Gly Ser Lys Gly Val Val Phe Gly
115 120 125
Pro Ile Lys Asp Met Ala His Ile Ser His Gly Pro Val Gly Cys Gly
130 135 140
Gln Tyr Ser Ala Ala Ala Ala Ala Asn Tyr Tyr Thr Gly Val Ser Gly
145 150 155 160
Val Asp Ser Phe Gly Thr Leu Asn Phe Thr Ser Asp Phe Gln Glu Arg
165 170 175
Asp Ile Val Phe Gly Gly Asp Lys Lys Leu Ser Lys Leu Ile Glu Glu
180 185 190
Met Glu Leu Leu Phe Pro Leu Thr Lys Gly Ile Thr Ile Gln Ser Glu
195 200 205
Cys Pro Val Gly Leu Ile Gly Asp Asp Ile Ser Ala Val Ala Asn Ala
210 215 220
Ser Ser Lys Ala Leu Asp Lys Pro Val Ile Pro Val Arg Cys Glu Gly
225 230 235 240
Phe Arg Gly Val Ser Gln Ser Leu Gly His His Ile Ala Asn Asp Val
245 250 255
Val Arg Asp Trp Ile Leu Asn Asn Arg Glu Gly Gln Pro Phe Glu Thr
260 265 270
Thr Pro Tyr Asp Val Ala Ile Ile Gly Asp Tyr Asn Ile Gly Gly Asp
275 280 285
Ala Trp Ala Ser Arg Ile Leu Leu Glu Glu Met Gly Leu Arg Val Val
290 295 300
Ala Gln Trp Ser Gly Asp Gly Thr Leu Val Glu Met Glu Asn Thr Pro
305 310 315 320
Phe Val Lys Leu Asn Leu Val His Cys Tyr Arg Ser Met Asn Tyr Ile
325 330 335
Ala Arg His Met Glu Glu Lys His Gln Ile Pro Trp Met Glu Tyr Asn
340 345 350
Phe Phe Gly Pro Thr Lys Ile Ala Glu Ser Leu Arg Lys Ile Ala Asp
355 360 365
Gln Phe Asp Asp Thr Ile Arg Ala Asn Ala Glu Ala Val Ile Ala Arg
370 375 380
Tyr Glu Gly Gln Met Ala Ala Ile Ile Ala Lys Tyr Arg Pro Arg Leu
385 390 395 400
Glu Gly Arg Lys Val Leu Leu Tyr Met Gly Gly Leu Arg Pro Arg His
405 410 415
Val Ile Gly Ala Tyr Glu Asp Leu Gly Met Glu Ile Ile Ala Ala Gly
420 425 430
Tyr Glu Phe Ala His Asn Asp Asp Tyr Asp Arg Thr Leu Pro Asp Leu
435 440 445
Lys Glu Gly Thr Leu Leu Phe Asp Asp Ala Ser Ser Tyr Glu Leu Glu
450 455 460
Ala Phe Val Lys Ala Leu Lys Pro Asp Leu Ile Gly Ser Gly Ile Lys
465 470 475 480
Glu Lys Tyr Ile Phe Gln Lys Met Gly Val Pro Phe Arg Gln Met His
485 490 495
Ser Trp Asp Tyr Ser Gly Pro Tyr His Gly Tyr Asp Gly Phe Ala Ile
500 505 510
Phe Ala Arg Asp Met Asp Met Thr Leu Asn Asn Pro Ala Trp Asn Glu
515 520 525
Leu Thr Ala Pro Trp Leu Lys Ser Gly Gly Tyr Pro Tyr Asp Val Pro
530 535 540
Asp Tyr Ala
545
<210> 134
<211> 547
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-FA
51::NifH::HA, кодированная SN100. Аминокислоты 1-54 соответствуют
MTP-FA 51 с GG на ее C-конце, аминокислоты 55-536 соответствуют
аминокислотам NifD K.oxytoca, содержащим замену аланина
<400> 134
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly Met Met Thr Asn Ala Thr Gly Glu Arg Asn
50 55 60
Leu Ala Leu Ile Gln Glu Val Leu Glu Val Phe Pro Glu Thr Ala Arg
65 70 75 80
Lys Glu Arg Arg Lys His Met Met Val Ser Asp Pro Glu Met Glu Ser
85 90 95
Val Gly Lys Cys Ile Ile Ser Asn Arg Lys Ser Gln Pro Gly Val Met
100 105 110
Thr Val Arg Gly Cys Ala Tyr Ala Gly Ser Lys Gly Val Val Phe Gly
115 120 125
Pro Ile Lys Asp Met Ala His Ile Ser His Gly Pro Val Gly Cys Gly
130 135 140
Gln Tyr Ser Arg Ala Gly Arg Arg Ala Ala Ala Ala Ala Val Ser Gly
145 150 155 160
Val Asp Ser Phe Gly Thr Leu Asn Phe Thr Ser Asp Phe Gln Glu Arg
165 170 175
Asp Ile Val Phe Gly Gly Asp Lys Lys Leu Ser Lys Leu Ile Glu Glu
180 185 190
Met Glu Leu Leu Phe Pro Leu Thr Lys Gly Ile Thr Ile Gln Ser Glu
195 200 205
Cys Pro Val Gly Leu Ile Gly Asp Asp Ile Ser Ala Val Ala Asn Ala
210 215 220
Ser Ser Lys Ala Leu Asp Lys Pro Val Ile Pro Val Arg Cys Glu Gly
225 230 235 240
Phe Arg Gly Val Ser Gln Ser Leu Gly His His Ile Ala Asn Asp Val
245 250 255
Val Arg Asp Trp Ile Leu Asn Asn Arg Glu Gly Gln Pro Phe Glu Thr
260 265 270
Thr Pro Tyr Asp Val Ala Ile Ile Gly Asp Tyr Asn Ile Gly Gly Asp
275 280 285
Ala Trp Ala Ser Arg Ile Leu Leu Glu Glu Met Gly Leu Arg Val Val
290 295 300
Ala Gln Trp Ser Gly Asp Gly Thr Leu Val Glu Met Glu Asn Thr Pro
305 310 315 320
Phe Val Lys Leu Asn Leu Val His Cys Tyr Arg Ser Met Asn Tyr Ile
325 330 335
Ala Arg His Met Glu Glu Lys His Gln Ile Pro Trp Met Glu Tyr Asn
340 345 350
Phe Phe Gly Pro Thr Lys Ile Ala Glu Ser Leu Arg Lys Ile Ala Asp
355 360 365
Gln Phe Asp Asp Thr Ile Arg Ala Asn Ala Glu Ala Val Ile Ala Arg
370 375 380
Tyr Glu Gly Gln Met Ala Ala Ile Ile Ala Lys Tyr Arg Pro Arg Leu
385 390 395 400
Glu Gly Arg Lys Val Leu Leu Tyr Met Gly Gly Leu Arg Pro Arg His
405 410 415
Val Ile Gly Ala Tyr Glu Asp Leu Gly Met Glu Ile Ile Ala Ala Gly
420 425 430
Tyr Glu Phe Ala His Asn Asp Asp Tyr Asp Arg Thr Leu Pro Asp Leu
435 440 445
Lys Glu Gly Thr Leu Leu Phe Asp Asp Ala Ser Ser Tyr Glu Leu Glu
450 455 460
Ala Phe Val Lys Ala Leu Lys Pro Asp Leu Ile Gly Ser Gly Ile Lys
465 470 475 480
Glu Lys Tyr Ile Phe Gln Lys Met Gly Val Pro Phe Arg Gln Met His
485 490 495
Ser Trp Asp Tyr Ser Gly Pro Tyr His Gly Tyr Asp Gly Phe Ala Ile
500 505 510
Phe Ala Arg Asp Met Asp Met Thr Leu Asn Asn Pro Ala Trp Asn Glu
515 520 525
Leu Thr Ala Pro Trp Leu Lys Ser Gly Gly Tyr Pro Tyr Asp Val Pro
530 535 540
Asp Tyr Ala
545
<210> 135
<211> 167
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-Su9::NifW,
кодированная SN104. Аминокислоты 1-70 соответствуют MTP-Su9 с
с GG на ее C-конце, аминокислоты 71-158 соответствуют NifW K. oxytoca
(SEQ ID NO:17) с ее инициатором Met, и амино
<400> 135
Met Ala Ser Thr Arg Val Leu Ala Ser Arg Leu Ala Ser Gln Met Ala
1. 5 10 15
Ala Ser Ala Lys Val Ala Arg Pro Ala Val Arg Val Ala Gln Val Ser
20 25 30
Lys Arg Thr Ile Gln Thr Gly Ser Pro Leu Gln Thr Leu Lys Arg Thr
35 40 45
Gln Met Thr Ser Ile Val Asn Ala Thr Thr Arg Gln Ala Phe Gln Lys
50 55 60
Arg Ala Tyr Ser Gly Gly Met Met Glu Trp Phe Tyr Gln Ile Pro Gly
65 70 75 80
Val Asp Glu Leu Arg Ser Ala Glu Ser Phe Phe Gln Phe Phe Ala Val
85 90 95
Pro Tyr Gln Pro Glu Leu Leu Gly Arg Cys Ser Leu Pro Val Leu Ala
100 105 110
Thr Phe His Arg Lys Leu Arg Ala Glu Val Pro Leu Gln Asn Arg Leu
115 120 125
Glu Asp Asn Asp Arg Ala Pro Trp Leu Leu Ala Arg Arg Leu Leu Ala
130 135 140
Glu Ser Tyr Gln Gln Gln Phe Gln Glu Ser Gly Thr Gly Gly Tyr Pro
145 150 155 160
Tyr Asp Val Pro Asp Tyr Ala
165
<210> 136
<211> 547
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-FA
51::NifH::HA, кодированная SN114. Аминокислоты 1-54 соответствуют
MTP-FA 51 с GG на ее C-конце, аминокислоты 55-536 соответствуют
NifD K.oxytoca, содержащие Y100Q мутации по типу замены в амино
<400> 136
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly Met Met Thr Asn Ala Thr Gly Glu Arg Asn
50 55 60
Leu Ala Leu Ile Gln Glu Val Leu Glu Val Phe Pro Glu Thr Ala Arg
65 70 75 80
Lys Glu Arg Arg Lys His Met Met Val Ser Asp Pro Glu Met Glu Ser
85 90 95
Val Gly Lys Cys Ile Ile Ser Asn Arg Lys Ser Gln Pro Gly Val Met
100 105 110
Thr Val Arg Gly Cys Ala Tyr Ala Gly Ser Lys Gly Val Val Phe Gly
115 120 125
Pro Ile Lys Asp Met Ala His Ile Ser His Gly Pro Val Gly Cys Gly
130 135 140
Gln Tyr Ser Arg Ala Gly Arg Arg Asn Gln Tyr Thr Gly Val Ser Gly
145 150 155 160
Val Asp Ser Phe Gly Thr Leu Asn Phe Thr Ser Asp Phe Gln Glu Arg
165 170 175
Asp Ile Val Phe Gly Gly Asp Lys Lys Leu Ser Lys Leu Ile Glu Glu
180 185 190
Met Glu Leu Leu Phe Pro Leu Thr Lys Gly Ile Thr Ile Gln Ser Glu
195 200 205
Cys Pro Val Gly Leu Ile Gly Asp Asp Ile Ser Ala Val Ala Asn Ala
210 215 220
Ser Ser Lys Ala Leu Asp Lys Pro Val Ile Pro Val Arg Cys Glu Gly
225 230 235 240
Phe Arg Gly Val Ser Gln Ser Leu Gly His His Ile Ala Asn Asp Val
245 250 255
Val Arg Asp Trp Ile Leu Asn Asn Arg Glu Gly Gln Pro Phe Glu Thr
260 265 270
Thr Pro Tyr Asp Val Ala Ile Ile Gly Asp Tyr Asn Ile Gly Gly Asp
275 280 285
Ala Trp Ala Ser Arg Ile Leu Leu Glu Glu Met Gly Leu Arg Val Val
290 295 300
Ala Gln Trp Ser Gly Asp Gly Thr Leu Val Glu Met Glu Asn Thr Pro
305 310 315 320
Phe Val Lys Leu Asn Leu Val His Cys Tyr Arg Ser Met Asn Tyr Ile
325 330 335
Ala Arg His Met Glu Glu Lys His Gln Ile Pro Trp Met Glu Tyr Asn
340 345 350
Phe Phe Gly Pro Thr Lys Ile Ala Glu Ser Leu Arg Lys Ile Ala Asp
355 360 365
Gln Phe Asp Asp Thr Ile Arg Ala Asn Ala Glu Ala Val Ile Ala Arg
370 375 380
Tyr Glu Gly Gln Met Ala Ala Ile Ile Ala Lys Tyr Arg Pro Arg Leu
385 390 395 400
Glu Gly Arg Lys Val Leu Leu Tyr Met Gly Gly Leu Arg Pro Arg His
405 410 415
Val Ile Gly Ala Tyr Glu Asp Leu Gly Met Glu Ile Ile Ala Ala Gly
420 425 430
Tyr Glu Phe Ala His Asn Asp Asp Tyr Asp Arg Thr Leu Pro Asp Leu
435 440 445
Lys Glu Gly Thr Leu Leu Phe Asp Asp Ala Ser Ser Tyr Glu Leu Glu
450 455 460
Ala Phe Val Lys Ala Leu Lys Pro Asp Leu Ile Gly Ser Gly Ile Lys
465 470 475 480
Glu Lys Tyr Ile Phe Gln Lys Met Gly Val Pro Phe Arg Gln Met His
485 490 495
Ser Trp Asp Tyr Ser Gly Pro Tyr His Gly Tyr Asp Gly Phe Ala Ile
500 505 510
Phe Ala Arg Asp Met Asp Met Thr Leu Asn Asn Pro Ala Trp Asn Glu
515 520 525
Leu Thr Ala Pro Trp Leu Lys Ser Gly Gly Tyr Pro Tyr Asp Val Pro
530 535 540
Asp Tyr Ala
545
<210> 137
<211> 241
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-FA
51::NifF::HA, кодированная SN138. Аминокислоты 1-54 соответствуют
MTP-FA 51 с GG, аминокислоты 55-230 соответствуют NifF
K. oxytoca (SEQ ID NO:6), а аминокислоты 231-241 включают эпитоп HA.
<400> 137
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly Met Ala Asn Ile Gly Ile Phe Phe Gly Thr
50 55 60
Asp Thr Gly Lys Thr Arg Lys Ile Ala Lys Met Ile His Lys Gln Leu
65 70 75 80
Gly Glu Leu Ala Asp Ala Pro Val Asn Ile Asn Arg Thr Thr Leu Asp
85 90 95
Asp Phe Met Ala Tyr Pro Val Leu Leu Leu Gly Thr Pro Thr Leu Gly
100 105 110
Asp Gly Gln Leu Pro Gly Leu Glu Ala Gly Cys Glu Ser Glu Ser Trp
115 120 125
Ser Glu Phe Ile Ser Gly Leu Asp Asp Ala Ser Leu Lys Gly Lys Thr
130 135 140
Val Ala Leu Phe Gly Leu Gly Asp Gln Arg Gly Tyr Pro Asp Asn Phe
145 150 155 160
Val Ser Gly Met Arg Pro Leu Phe Asp Ala Leu Ser Ala Arg Gly Ala
165 170 175
Gln Met Ile Gly Ser Trp Pro Asn Glu Gly Tyr Glu Phe Ser Ala Ser
180 185 190
Ser Ala Leu Glu Gly Asp Arg Phe Val Gly Leu Val Leu Asp Gln Asp
195 200 205
Asn Gln Phe Asp Gln Thr Glu Ala Arg Leu Ala Ser Trp Leu Glu Glu
210 215 220
Ile Lys Arg Thr Val Leu Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr
225 230 235 240
Ala
<210> 138
<211> 1236
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-FA
51::NifJ::HA, кодированная SN139. Аминокислоты 1-54 соответствуют
MTP-FA 51 с GG, аминокислоты 55-1225 соответствуют NifJ
K. oxytoca (SEQ ID NO:7), а аминокислоты 1226-1236 включают эпитоп HA.
<400> 138
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly Met Ser Gly Lys Met Lys Thr Met Asp Gly
50 55 60
Asn Ala Ala Ala Ala Trp Ile Ser Tyr Ala Phe Thr Glu Val Ala Ala
65 70 75 80
Ile Tyr Pro Ile Thr Pro Ser Thr Pro Met Ala Glu Asn Val Asp Glu
85 90 95
Trp Ala Ala Gln Gly Lys Lys Asn Leu Phe Gly Gln Pro Val Arg Leu
100 105 110
Met Glu Met Gln Ser Glu Ala Gly Ala Ala Gly Ala Val His Gly Ala
115 120 125
Leu Gln Ala Gly Ala Leu Thr Thr Thr Tyr Thr Ala Ser Gln Gly Leu
130 135 140
Leu Leu Met Ile Pro Asn Met Tyr Lys Ile Ala Gly Glu Leu Leu Pro
145 150 155 160
Gly Val Phe His Val Ser Ala Arg Ala Leu Ala Thr Asn Ser Leu Asn
165 170 175
Ile Phe Gly Asp His Gln Asp Val Met Ala Val Arg Gln Thr Gly Cys
180 185 190
Ala Met Leu Ala Glu Asn Asn Val Gln Gln Val Met Asp Leu Ser Ala
195 200 205
Val Ala His Leu Ala Ala Ile Lys Gly Arg Ile Pro Phe Val Asn Phe
210 215 220
Phe Asp Gly Phe Arg Thr Ser His Glu Ile Gln Lys Ile Glu Val Leu
225 230 235 240
Glu Tyr Glu Gln Leu Ala Thr Leu Leu Asp Arg Pro Ala Leu Asp Ser
245 250 255
Phe Arg Arg Asn Ala Leu His Pro Asp His Pro Val Ile Arg Gly Thr
260 265 270
Ala Gln Asn Pro Asp Ile Tyr Phe Gln Glu Arg Glu Ala Gly Asn Arg
275 280 285
Phe Tyr Gln Ala Leu Pro Asp Ile Val Glu Ser Tyr Met Thr Gln Ile
290 295 300
Ser Ala Leu Thr Gly Arg Glu Tyr His Leu Phe Asn Tyr Thr Gly Ala
305 310 315 320
Ala Asp Ala Glu Arg Val Ile Ile Ala Met Gly Ser Val Cys Asp Thr
325 330 335
Val Gln Glu Val Val Asp Thr Leu Asn Ala Ala Gly Glu Lys Val Gly
340 345 350
Leu Leu Ser Val His Leu Phe Arg Pro Phe Ser Leu Ala His Phe Phe
355 360 365
Ala Gln Leu Pro Lys Thr Val Gln Arg Ile Ala Val Leu Asp Arg Thr
370 375 380
Lys Glu Pro Gly Ala Gln Ala Glu Pro Leu Cys Leu Asp Val Lys Asn
385 390 395 400
Ala Phe Tyr His His Asp Asp Ala Pro Leu Ile Val Gly Gly Arg Tyr
405 410 415
Ala Leu Gly Gly Lys Asp Val Leu Pro Asn Asp Ile Ala Ala Val Phe
420 425 430
Asp Asn Leu Asn Lys Pro Leu Pro Met Asp Gly Phe Thr Leu Gly Ile
435 440 445
Val Asp Asp Val Thr Phe Thr Ser Leu Pro Pro Arg Gln Gln Thr Leu
450 455 460
Ala Val Ser His Asp Gly Ile Thr Ala Cys Lys Phe Trp Gly Met Gly
465 470 475 480
Ser Asp Gly Thr Val Gly Ala Asn Lys Ser Ala Ile Lys Ile Ile Gly
485 490 495
Asp Lys Thr Pro Leu Tyr Ala Gln Ala Tyr Phe Ser Tyr Asp Ser Lys
500 505 510
Lys Ser Gly Gly Ile Thr Val Ser His Leu Arg Phe Gly Asp Arg Pro
515 520 525
Ile Asn Ser Pro Tyr Leu Ile His Arg Ala Asp Phe Ile Ser Cys Ser
530 535 540
Gln Gln Ser Tyr Val Glu Arg Tyr Asp Leu Leu Asp Gly Leu Lys Pro
545 550 555 560
Gly Gly Thr Phe Leu Leu Asn Cys Ser Trp Ser Asp Ala Glu Leu Glu
565 570 575
Gln His Leu Pro Val Gly Phe Lys Arg Tyr Leu Ala Arg Glu Asn Ile
580 585 590
His Phe Tyr Thr Leu Asn Ala Val Asp Ile Ala Arg Glu Leu Gly Leu
595 600 605
Gly Gly Arg Phe Asn Met Leu Met Gln Ala Ala Phe Phe Lys Leu Ala
610 615 620
Ala Ile Ile Asp Pro Gln Thr Ala Ala Asp Tyr Leu Lys Gln Ala Val
625 630 635 640
Glu Lys Ser Tyr Gly Ser Lys Gly Ala Ala Val Ile Glu Met Asn Gln
645 650 655
Arg Ala Ile Glu Leu Gly Met Ala Ser Leu His Gln Val Thr Ile Pro
660 665 670
Ala His Trp Ala Thr Leu Asp Glu Pro Ala Ala Gln Ala Ser Ala Met
675 680 685
Met Pro Asp Phe Ile Arg Asp Ile Leu Gln Pro Met Asn Arg Gln Cys
690 695 700
Gly Asp Gln Leu Pro Val Ser Ala Phe Val Gly Met Glu Asp Gly Thr
705 710 715 720
Phe Pro Ser Gly Thr Ala Ala Trp Glu Lys Arg Gly Ile Ala Leu Glu
725 730 735
Val Pro Val Trp Gln Pro Glu Gly Cys Thr Gln Cys Asn Gln Cys Ala
740 745 750
Phe Ile Cys Pro His Ala Ala Ile Arg Pro Ala Leu Leu Asn Gly Glu
755 760 765
Glu His Asp Ala Ala Pro Val Gly Leu Leu Ser Lys Pro Ala Gln Gly
770 775 780
Ala Lys Glu Tyr His Tyr His Leu Ala Ile Ser Pro Leu Asp Cys Ser
785 790 795 800
Gly Cys Gly Asn Cys Val Asp Ile Cys Pro Ala Arg Gly Lys Ala Leu
805 810 815
Lys Met Gln Ser Leu Asp Ser Gln Arg Gln Met Ala Pro Val Trp Asp
820 825 830
Tyr Ala Leu Ala Leu Thr Pro Lys Ser Asn Pro Phe Arg Lys Thr Thr
835 840 845
Val Lys Gly Ser Gln Phe Glu Thr Pro Leu Leu Glu Phe Ser Gly Ala
850 855 860
Cys Ala Gly Cys Gly Glu Thr Pro Tyr Ala Arg Leu Ile Thr Gln Leu
865 870 875 880
Phe Gly Asp Arg Met Leu Ile Ala Asn Ala Thr Gly Cys Ser Ser Ile
885 890 895
Trp Gly Ala Ser Ala Pro Ser Ile Pro Tyr Thr Thr Asn His Arg Gly
900 905 910
His Gly Pro Ala Trp Ala Asn Ser Leu Phe Glu Asp Asn Ala Glu Phe
915 920 925
Gly Leu Gly Met Met Leu Gly Gly Gln Ala Val Arg Gln Gln Ile Ala
930 935 940
Asp Asp Met Thr Ala Ala Leu Ala Leu Pro Val Ser Asp Glu Leu Ser
945 950 955 960
Asp Ala Met Arg Gln Trp Leu Ala Lys Gln Asp Glu Gly Glu Gly Thr
965 970 975
Arg Glu Arg Ala Asp Arg Leu Ser Glu Arg Leu Ala Ala Glu Lys Glu
980 985 990
Gly Val Pro Leu Leu Glu Gln Leu Trp Gln Asn Arg Asp Tyr Phe Val
995 1000 1005
Arg Arg Ser Gln Trp Ile Phe Gly Gly Asp Gly Trp Ala Tyr Asp
1010 1015 1020
Ile Gly Phe Gly Gly Leu Asp His Val Leu Ala Ser Gly Glu Asp
1025 1030 1035
Val Asn Ile Leu Val Phe Asp Thr Glu Val Tyr Ser Asn Thr Gly
1040 1045 1050
Gly Gln Ser Ser Lys Ser Thr Pro Val Ala Ala Ile Ala Lys Phe
1055 1060 1065
Ala Ala Gln Gly Lys Arg Thr Arg Lys Lys Asp Leu Gly Met Met
1070 1075 1080
Ala Met Ser Tyr Gly Asn Val Tyr Val Ala Gln Val Ala Met Gly
1085 1090 1095
Ala Asp Lys Asp Gln Thr Leu Arg Ala Ile Ala Glu Ala Glu Ala
1100 1105 1110
Trp Pro Gly Pro Ser Leu Val Ile Ala Tyr Ala Ala Cys Ile Asn
1115 1120 1125
His Gly Leu Lys Ala Gly Met Arg Cys Ser Gln Arg Glu Ala Lys
1130 1135 1140
Arg Ala Val Glu Ala Gly Tyr Trp His Leu Trp Arg Tyr His Pro
1145 1150 1155
Gln Arg Glu Ala Glu Gly Lys Thr Pro Phe Met Leu Asp Ser Glu
1160 1165 1170
Glu Pro Glu Glu Ser Phe Arg Asp Phe Leu Leu Gly Glu Val Arg
1175 1180 1185
Tyr Ala Ser Leu His Lys Thr Thr Pro His Leu Ala Asp Ala Leu
1190 1195 1200
Phe Ser Arg Thr Glu Glu Asp Ala Arg Ala Arg Phe Ala Gln Tyr
1205 1210 1215
Arg Arg Leu Ala Gly Glu Glu Gly Gly Tyr Pro Tyr Asp Val Pro
1220 1225 1230
Asp Tyr Ala
1235
<210> 139
<211> 584
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-FA
51::HA::NifK, кодированная SN140. Аминокислоты 1-53 соответствуют
MTP-FA 51 с GG, аминокислоты 54-64 включают эпитоп HA, а аминокислоты
65-584 соответствуют NifK K. Oxytoca (SEQ ID NO:3) с
<400> 139
Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro Ser
1. 5 10 15
Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser Asp
20 25 30
Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val Val
35 40 45
Arg Asn Arg Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Gly Gly
50 55 60
Met Ser Gln Thr Ile Asp Lys Ile Asn Ser Cys Tyr Pro Leu Phe Glu
65 70 75 80
Gln Asp Glu Tyr Gln Glu Leu Phe Arg Asn Lys Arg Gln Leu Glu Glu
85 90 95
Ala His Asp Ala Gln Arg Val Gln Glu Val Phe Ala Trp Thr Thr Thr
100 105 110
Ala Glu Tyr Glu Ala Leu Asn Phe Gln Arg Glu Ala Leu Thr Val Asp
115 120 125
Pro Ala Lys Ala Cys Gln Pro Leu Gly Ala Val Leu Cys Ser Leu Gly
130 135 140
Phe Ala Asn Thr Leu Pro Tyr Val His Gly Ser Gln Gly Cys Val Ala
145 150 155 160
Tyr Phe Arg Thr Tyr Phe Asn Arg His Phe Lys Glu Pro Ile Ala Cys
165 170 175
Val Ser Asp Ser Met Thr Glu Asp Ala Ala Val Phe Gly Gly Asn Asn
180 185 190
Asn Met Asn Leu Gly Leu Gln Asn Ala Ser Ala Leu Tyr Lys Pro Glu
195 200 205
Ile Ile Ala Val Ser Thr Thr Cys Met Ala Glu Val Ile Gly Asp Asp
210 215 220
Leu Gln Ala Phe Ile Ala Asn Ala Lys Lys Asp Gly Phe Val Asp Ser
225 230 235 240
Ser Ile Ala Val Pro His Ala His Thr Pro Ser Phe Ile Gly Ser His
245 250 255
Val Thr Gly Trp Asp Asn Met Phe Glu Gly Phe Ala Lys Thr Phe Thr
260 265 270
Ala Asp Tyr Gln Gly Gln Pro Gly Lys Leu Pro Lys Leu Asn Leu Val
275 280 285
Thr Gly Phe Glu Thr Tyr Leu Gly Asn Phe Arg Val Leu Lys Arg Met
290 295 300
Met Glu Gln Met Ala Val Pro Cys Ser Leu Leu Ser Asp Pro Ser Glu
305 310 315 320
Val Leu Asp Thr Pro Ala Asp Gly His Tyr Arg Met Tyr Ser Gly Gly
325 330 335
Thr Thr Gln Gln Glu Met Lys Glu Ala Pro Asp Ala Ile Asp Thr Leu
340 345 350
Leu Leu Gln Pro Trp Gln Leu Leu Lys Ser Lys Lys Val Val Gln Glu
355 360 365
Met Trp Asn Gln Pro Ala Thr Glu Val Ala Ile Pro Leu Gly Leu Ala
370 375 380
Ala Thr Asp Glu Leu Leu Met Thr Val Ser Gln Leu Ser Gly Lys Pro
385 390 395 400
Ile Ala Asp Ala Leu Thr Leu Glu Arg Gly Arg Leu Val Asp Met Met
405 410 415
Leu Asp Ser His Thr Trp Leu His Gly Lys Lys Phe Gly Leu Tyr Gly
420 425 430
Asp Pro Asp Phe Val Met Gly Leu Thr Arg Phe Leu Leu Glu Leu Gly
435 440 445
Cys Glu Pro Thr Val Ile Leu Ser His Asn Ala Asn Lys Arg Trp Gln
450 455 460
Lys Ala Met Asn Lys Met Leu Asp Ala Ser Pro Tyr Gly Arg Asp Ser
465 470 475 480
Glu Val Phe Ile Asn Cys Asp Leu Trp His Phe Arg Ser Leu Met Phe
485 490 495
Thr Arg Gln Pro Asp Phe Met Ile Gly Asn Ser Tyr Gly Lys Phe Ile
500 505 510
Gln Arg Asp Thr Leu Ala Lys Gly Lys Ala Phe Glu Val Pro Leu Ile
515 520 525
Arg Leu Gly Phe Pro Leu Phe Asp Arg His His Leu His Arg Gln Thr
530 535 540
Thr Trp Gly Tyr Glu Gly Ala Met Asn Ile Val Thr Thr Leu Val Asn
545 550 555 560
Ala Val Leu Glu Lys Leu Asp Ser Asp Thr Ser Gln Leu Gly Lys Thr
565 570 575
Asp Tyr Ser Phe Asp Leu Val Arg
580
<210> 140
<211> 232
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-FA
51::NifQ::HA,кодированная SN141. Аминокислоты 1-54 соответствуют
MTP-FAγ51 с GG, аминокислоты 55-221 соответствуют NifQ K. oxytoca
(SEQ ID NO:10), а аминокислоты 222-232 включают эпитоп HA.
<400> 140
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly Met Pro Pro Leu Asp Trp Leu Arg Arg Leu
50 55 60
Trp Leu Leu Tyr His Ala Gly Lys Gly Ser Phe Pro Leu Arg Met Gly
65 70 75 80
Leu Ser Pro Arg Asp Trp Gln Ala Leu Arg Arg Arg Leu Gly Glu Val
85 90 95
Glu Thr Pro Leu Asp Gly Glu Thr Leu Thr Arg Arg Arg Leu Met Ala
100 105 110
Glu Leu Asn Ala Thr Arg Glu Glu Glu Arg Gln Gln Leu Gly Ala Trp
115 120 125
Leu Ala Gly Trp Met Gln Gln Asp Ala Gly Pro Met Ala Gln Ile Ile
130 135 140
Ala Glu Val Ser Leu Ala Phe Asn His Leu Trp Gln Asp Leu Gly Leu
145 150 155 160
Ala Ser Arg Ala Glu Leu Arg Leu Leu Met Ser Asp Cys Phe Pro Gln
165 170 175
Leu Val Val Met Asn Glu His Asn Met Arg Trp Lys Lys Phe Phe Tyr
180 185 190
Arg Gln Arg Cys Leu Leu Gln Gln Gly Glu Val Ile Cys Arg Ser Pro
195 200 205
Ser Cys Asp Glu Cys Trp Glu Arg Ser Ala Cys Phe Glu Gly Gly Tyr
210 215 220
Pro Tyr Asp Val Pro Asp Tyr Ala
225 230
<210> 141
<211> 446
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-FA
51::NifV::HA, кодированная SN142. Аминокислоты 1-54 соответствуют
MTPFA 51 с GG, аминокислоты 55-435 соответствуют NifV K. oxytoca
(SEQ ID NO:13), а аминокислоты 436-446 включают эпитоп HA.
<400> 141
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly Met Glu Arg Val Leu Ile Asn Asp Thr Thr
50 55 60
Leu Arg Asp Gly Glu Gln Ser Pro Gly Val Ala Phe Arg Thr Ser Glu
65 70 75 80
Lys Val Ala Ile Ala Glu Ala Leu Tyr Ala Ala Gly Ile Thr Ala Met
85 90 95
Glu Val Gly Thr Pro Ala Met Gly Asp Glu Glu Ile Ala Arg Ile Gln
100 105 110
Leu Val Arg Arg Gln Leu Pro Asp Ala Thr Leu Met Thr Trp Cys Arg
115 120 125
Met Asn Ala Leu Glu Ile Arg Gln Ser Ala Asp Leu Gly Ile Asp Trp
130 135 140
Val Asp Ile Ser Ile Pro Ala Ser Asp Lys Leu Arg Gln Tyr Lys Leu
145 150 155 160
Arg Glu Pro Leu Ala Val Leu Leu Glu Arg Leu Ala Met Phe Ile His
165 170 175
Leu Ala His Thr Leu Gly Leu Lys Val Cys Ile Gly Cys Glu Asp Ala
180 185 190
Ser Arg Ala Ser Gly Gln Thr Leu Arg Ala Ile Ala Glu Val Ala Gln
195 200 205
Asn Ala Pro Ala Ala Arg Leu Arg Tyr Ala Asp Thr Val Gly Leu Leu
210 215 220
Asp Pro Phe Thr Thr Ala Ala Gln Ile Ser Ala Leu Arg Asp Val Trp
225 230 235 240
Ser Gly Glu Ile Glu Met His Ala His Asn Asp Leu Gly Met Ala Thr
245 250 255
Ala Asn Thr Leu Ala Ala Val Ser Ala Gly Ala Thr Ser Val Asn Thr
260 265 270
Thr Val Leu Gly Leu Gly Glu Arg Ala Gly Asn Ala Ala Ala Trp Lys
275 280 285
Pro Ser Ala Leu Gly Leu Glu Arg Cys Leu Gly Val Glu Thr Gly Val
290 295 300
His Phe Ser Ala Leu Pro Ala Leu Cys Gln Arg Val Ala Glu Ala Ala
305 310 315 320
Gln Arg Ala Ile Asp Pro Gln Gln Pro Leu Val Gly Glu Leu Val Phe
325 330 335
Thr His Glu Ser Gly Val His Val Ala Ala Leu Leu Arg Asp Ser Glu
340 345 350
Ser Tyr Gln Ser Ile Ala Pro Ser Leu Met Gly Arg Ser Tyr Arg Leu
355 360 365
Val Leu Gly Lys His Ser Gly Arg Gln Ala Val Asn Gly Val Phe Asp
370 375 380
Gln Met Gly Tyr His Leu Asn Ala Ala Gln Ile Asn Gln Leu Leu Pro
385 390 395 400
Ala Ile Arg Arg Phe Ala Glu Asn Trp Lys Arg Ser Pro Lys Asp Tyr
405 410 415
Glu Leu Val Ala Ile Tyr Asp Glu Leu Cys Gly Glu Ser Ala Leu Arg
420 425 430
Ala Arg Gly Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala
435 440 445
<210> 142
<211> 151
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-FA
51::NifW::HA, кодированная SN143. Аминокислоты 1-54 соответствуют
MTP-FA 51 с GG,аминокислоты 55-140 соответствуют NifW K. oxytoca
(SEQ ID NO:17), а аминокислоты 141-151 включают эпитоп HA.
<400> 142
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly Met Met Glu Trp Phe Tyr Gln Ile Pro Gly
50 55 60
Val Asp Glu Leu Arg Ser Ala Glu Ser Phe Phe Gln Phe Phe Ala Val
65 70 75 80
Pro Tyr Gln Pro Glu Leu Leu Gly Arg Cys Ser Leu Pro Val Leu Ala
85 90 95
Thr Phe His Arg Lys Leu Arg Ala Glu Val Pro Leu Gln Asn Arg Leu
100 105 110
Glu Asp Asn Asp Arg Ala Pro Trp Leu Leu Ala Arg Arg Leu Leu Ala
115 120 125
Glu Ser Tyr Gln Gln Gln Phe Gln Glu Ser Gly Thr Gly Gly Tyr Pro
130 135 140
Tyr Asp Val Pro Asp Tyr Ala
145 150
<210> 143
<211> 221
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-FA
51::NifX::HA, кодированная SN144. Аминокислоты 1-54 соответствуют
MTP-FA 51 с GG,аминокислоты 55-210 соответствуют NifX K. Oxytoca
(SEQ ID NO:14), а аминокислоты 211-221 включают эпитоп HA.
<400> 143
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly Met Pro Pro Ile Asn Arg Gln Phe Asp Met
50 55 60
Val His Ser Asp Glu Trp Ser Met Lys Val Ala Phe Ala Ser Ser Asp
65 70 75 80
Tyr Arg His Val Asp Gln His Phe Gly Ala Thr Pro Arg Leu Val Val
85 90 95
Tyr Gly Val Lys Ala Asp Arg Val Thr Leu Ile Arg Val Val Asp Phe
100 105 110
Ser Val Glu Asn Gly His Gln Thr Glu Lys Ile Ala Arg Arg Ile His
115 120 125
Ala Leu Glu Asp Cys Val Thr Leu Phe Cys Val Ala Ile Gly Asp Ala
130 135 140
Val Phe Arg Gln Leu Leu Gln Val Gly Val Arg Ala Glu Arg Val Pro
145 150 155 160
Ala Asp Thr Thr Ile Val Gly Leu Leu Gln Glu Ile Gln Leu Tyr Trp
165 170 175
Tyr Asp Lys Gly Gln Arg Lys Asn Gln Arg Gln Arg Asp Pro Glu Arg
180 185 190
Phe Thr Arg Leu Leu Gln Glu Gln Glu Trp His Gly Asp Pro Asp Pro
195 200 205
Arg Arg Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala
210 215 220
<210> 144
<211> 285
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-FA
51::NifY::HA, кодированная SN145. Аминокислоты 1-54 соответствуют MTP-
FA 51 с GG, аминокислоты 55-274 соответствуют NifY K. oxytoca
согласно работы Temme et al., (2012), а аминокислоты 275-285
<400> 144
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly Met Ser Asp Asn Asp Thr Leu Phe Trp Arg
50 55 60
Met Leu Ala Leu Phe Gln Ser Leu Pro Asp Leu Gln Pro Ala Gln Ile
65 70 75 80
Val Asp Trp Leu Ala Gln Glu Ser Gly Glu Thr Leu Thr Pro Glu Arg
85 90 95
Leu Ala Thr Leu Thr Gln Pro Gln Leu Ala Ala Ser Phe Pro Ser Ala
100 105 110
Thr Ala Val Met Ser Pro Ala Arg Trp Ser Arg Val Met Ala Ser Leu
115 120 125
Gln Gly Ala Leu Pro Ala His Leu Arg Ile Val Arg Pro Ala Gln Arg
130 135 140
Thr Pro Gln Leu Leu Ala Ala Phe Cys Ser Gln Asp Gly Leu Val Ile
145 150 155 160
Asn Gly His Phe Gly Gln Gly Arg Leu Phe Phe Ile Tyr Ala Phe Asp
165 170 175
Glu Gln Gly Gly Trp Leu Tyr Asp Leu Arg Arg Tyr Pro Ser Ala Pro
180 185 190
His Gln Gln Glu Ala Asn Glu Val Arg Ala Arg Leu Ile Glu Asp Cys
195 200 205
Gln Leu Leu Phe Cys Gln Glu Ile Gly Gly Pro Ala Ala Ala Arg Leu
210 215 220
Ile Arg His Arg Ile His Pro Met Lys Ala Gln Pro Gly Thr Thr Ile
225 230 235 240
Gln Ala Gln Cys Glu Ala Ile Asn Thr Leu Leu Ala Gly Arg Leu Pro
245 250 255
Pro Trp Leu Ala Lys Arg Leu Asn Arg Asp Asn Pro Leu Glu Glu Arg
260 265 270
Val Phe Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala
275 280 285
<210> 145
<211> 213
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MMTP-FA
51::NifZ::HA, кодированная SN146. Аминокислоты 1-54 соответствуют
MTP-FA 51 с GG,аминокислоты 55-202 соответствуют NifZ K. oxytoca
(SEQ ID NO:16), а аминокислоты 203-213 включают эпитоп HA.
<400> 145
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly Met Arg Pro Lys Phe Thr Phe Ser Glu Glu
50 55 60
Val Arg Val Val Arg Ala Ile Arg Asn Asp Gly Thr Val Ala Gly Phe
65 70 75 80
Ala Pro Gly Ala Leu Leu Val Arg Arg Gly Ser Thr Gly Phe Val Arg
85 90 95
Asp Trp Gly Val Phe Leu Gln Asp Gln Ile Ile Tyr Gln Ile His Phe
100 105 110
Pro Glu Thr Asp Arg Ile Ile Gly Cys Arg Glu Gln Glu Leu Ile Pro
115 120 125
Ile Thr Gln Pro Trp Leu Ala Gly Asn Leu Gln Tyr Arg Asp Ser Val
130 135 140
Thr Cys Gln Met Ala Leu Ala Val Asn Gly Asp Val Val Val Ser Ala
145 150 155 160
Gly Gln Arg Gly Arg Val Glu Ala Thr Asp Arg Gly Glu Leu Gly Asp
165 170 175
Ser Tyr Thr Val Asp Phe Ser Gly Arg Trp Phe Arg Val Pro Val Gln
180 185 190
Ala Ile Ala Leu Ile Glu Glu Arg Glu Glu Gly Gly Tyr Pro Tyr Asp
195 200 205
Val Pro Asp Tyr Ala
210
<210> 146
<211> 1085
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-FA
51::NifD(Y100Q)::линкер(HA)::NifK, кодированная SN159. Аминокислоты
1-54 соответствуют MTP-FA 51 с GG на ее C-конце, аминокислоты 55-536
соответствуют NifD K. oxytoca с заменой Y100Q, амино
<400> 146
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly Met Thr Asn Ala Thr Gly Glu Arg Asn Leu
50 55 60
Ala Leu Ile Gln Glu Val Leu Glu Val Phe Pro Glu Thr Ala Arg Lys
65 70 75 80
Glu Arg Arg Lys His Met Met Val Ser Asp Pro Glu Met Glu Ser Val
85 90 95
Gly Lys Cys Ile Ile Ser Asn Arg Lys Ser Gln Pro Gly Val Met Thr
100 105 110
Val Arg Gly Cys Ala Tyr Ala Gly Ser Lys Gly Val Val Phe Gly Pro
115 120 125
Ile Lys Asp Met Ala His Ile Ser His Gly Pro Val Gly Cys Gly Gln
130 135 140
Tyr Ser Arg Ala Gly Arg Arg Asn Gln Tyr Thr Gly Val Ser Gly Val
145 150 155 160
Asp Ser Phe Gly Thr Leu Asn Phe Thr Ser Asp Phe Gln Glu Arg Asp
165 170 175
Ile Val Phe Gly Gly Asp Lys Lys Leu Ser Lys Leu Ile Glu Glu Met
180 185 190
Glu Leu Leu Phe Pro Leu Thr Lys Gly Ile Thr Ile Gln Ser Glu Cys
195 200 205
Pro Val Gly Leu Ile Gly Asp Asp Ile Ser Ala Val Ala Asn Ala Ser
210 215 220
Ser Lys Ala Leu Asp Lys Pro Val Ile Pro Val Arg Cys Glu Gly Phe
225 230 235 240
Arg Gly Val Ser Gln Ser Leu Gly His His Ile Ala Asn Asp Val Val
245 250 255
Arg Asp Trp Ile Leu Asn Asn Arg Glu Gly Gln Pro Phe Glu Thr Thr
260 265 270
Pro Tyr Asp Val Ala Ile Ile Gly Asp Tyr Asn Ile Gly Gly Asp Ala
275 280 285
Trp Ala Ser Arg Ile Leu Leu Glu Glu Met Gly Leu Arg Val Val Ala
290 295 300
Gln Trp Ser Gly Asp Gly Thr Leu Val Glu Met Glu Asn Thr Pro Phe
305 310 315 320
Val Lys Leu Asn Leu Val His Cys Tyr Arg Ser Met Asn Tyr Ile Ala
325 330 335
Arg His Met Glu Glu Lys His Gln Ile Pro Trp Met Glu Tyr Asn Phe
340 345 350
Phe Gly Pro Thr Lys Ile Ala Glu Ser Leu Arg Lys Ile Ala Asp Gln
355 360 365
Phe Asp Asp Thr Ile Arg Ala Asn Ala Glu Ala Val Ile Ala Arg Tyr
370 375 380
Glu Gly Gln Met Ala Ala Ile Ile Ala Lys Tyr Arg Pro Arg Leu Glu
385 390 395 400
Gly Arg Lys Val Leu Leu Tyr Met Gly Gly Leu Arg Pro Arg His Val
405 410 415
Ile Gly Ala Tyr Glu Asp Leu Gly Met Glu Ile Ile Ala Ala Gly Tyr
420 425 430
Glu Phe Ala His Asn Asp Asp Tyr Asp Arg Thr Leu Pro Asp Leu Lys
435 440 445
Glu Gly Thr Leu Leu Phe Asp Asp Ala Ser Ser Tyr Glu Leu Glu Ala
450 455 460
Phe Val Lys Ala Leu Lys Pro Asp Leu Ile Gly Ser Gly Ile Lys Glu
465 470 475 480
Lys Tyr Ile Phe Gln Lys Met Gly Val Pro Phe Arg Gln Met His Ser
485 490 495
Trp Asp Tyr Ser Gly Pro Tyr His Gly Tyr Asp Gly Phe Ala Ile Phe
500 505 510
Ala Arg Asp Met Asp Met Thr Leu Asn Asn Pro Ala Trp Asn Glu Leu
515 520 525
Thr Ala Pro Trp Leu Lys Ser Ala Ala Thr Pro Pro Pro Gly Ser Thr
530 535 540
Thr Thr Ala Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Thr Pro Pro Pro
545 550 555 560
Gly Ser Thr Thr Thr Ala Ser Gln Thr Ile Asp Lys Ile Asn Ser Cys
565 570 575
Tyr Pro Leu Phe Glu Gln Asp Glu Tyr Gln Glu Leu Phe Arg Asn Lys
580 585 590
Arg Gln Leu Glu Glu Ala His Asp Ala Gln Arg Val Gln Glu Val Phe
595 600 605
Ala Trp Thr Thr Thr Ala Glu Tyr Glu Ala Leu Asn Phe Gln Arg Glu
610 615 620
Ala Leu Thr Val Asp Pro Ala Lys Ala Cys Gln Pro Leu Gly Ala Val
625 630 635 640
Leu Cys Ser Leu Gly Phe Ala Asn Thr Leu Pro Tyr Val His Gly Ser
645 650 655
Gln Gly Cys Val Ala Tyr Phe Arg Thr Tyr Phe Asn Arg His Phe Lys
660 665 670
Glu Pro Ile Ala Cys Val Ser Asp Ser Met Thr Glu Asp Ala Ala Val
675 680 685
Phe Gly Gly Asn Asn Asn Met Asn Leu Gly Leu Gln Asn Ala Ser Ala
690 695 700
Leu Tyr Lys Pro Glu Ile Ile Ala Val Ser Thr Thr Cys Met Ala Glu
705 710 715 720
Val Ile Gly Asp Asp Leu Gln Ala Phe Ile Ala Asn Ala Lys Lys Asp
725 730 735
Gly Phe Val Asp Ser Ser Ile Ala Val Pro His Ala His Thr Pro Ser
740 745 750
Phe Ile Gly Ser His Val Thr Gly Trp Asp Asn Met Phe Glu Gly Phe
755 760 765
Ala Lys Thr Phe Thr Ala Asp Tyr Gln Gly Gln Pro Gly Lys Leu Pro
770 775 780
Lys Leu Asn Leu Val Thr Gly Phe Glu Thr Tyr Leu Gly Asn Phe Arg
785 790 795 800
Val Leu Lys Arg Met Met Glu Gln Met Ala Val Pro Cys Ser Leu Leu
805 810 815
Ser Asp Pro Ser Glu Val Leu Asp Thr Pro Ala Asp Gly His Tyr Arg
820 825 830
Met Tyr Ser Gly Gly Thr Thr Gln Gln Glu Met Lys Glu Ala Pro Asp
835 840 845
Ala Ile Asp Thr Leu Leu Leu Gln Pro Trp Gln Leu Leu Lys Ser Lys
850 855 860
Lys Val Val Gln Glu Met Trp Asn Gln Pro Ala Thr Glu Val Ala Ile
865 870 875 880
Pro Leu Gly Leu Ala Ala Thr Asp Glu Leu Leu Met Thr Val Ser Gln
885 890 895
Leu Ser Gly Lys Pro Ile Ala Asp Ala Leu Thr Leu Glu Arg Gly Arg
900 905 910
Leu Val Asp Met Met Leu Asp Ser His Thr Trp Leu His Gly Lys Lys
915 920 925
Phe Gly Leu Tyr Gly Asp Pro Asp Phe Val Met Gly Leu Thr Arg Phe
930 935 940
Leu Leu Glu Leu Gly Cys Glu Pro Thr Val Ile Leu Ser His Asn Ala
945 950 955 960
Asn Lys Arg Trp Gln Lys Ala Met Asn Lys Met Leu Asp Ala Ser Pro
965 970 975
Tyr Gly Arg Asp Ser Glu Val Phe Ile Asn Cys Asp Leu Trp His Phe
980 985 990
Arg Ser Leu Met Phe Thr Arg Gln Pro Asp Phe Met Ile Gly Asn Ser
995 1000 1005
Tyr Gly Lys Phe Ile Gln Arg Asp Thr Leu Ala Lys Gly Lys Ala
1010 1015 1020
Phe Glu Val Pro Leu Ile Arg Leu Gly Phe Pro Leu Phe Asp Arg
1025 1030 1035
His His Leu His Arg Gln Thr Thr Trp Gly Tyr Glu Gly Ala Met
1040 1045 1050
Asn Ile Val Thr Thr Leu Val Asn Ala Val Leu Glu Lys Leu Asp
1055 1060 1065
Ser Asp Thr Ser Gln Leu Gly Lys Thr Asp Tyr Ser Phe Asp Leu
1070 1075 1080
Val Arg
1085
<210> 147
<211> 533
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-FA
51::NifB::HA, кодированная SN192. Аминокислоты 1-54 соответствуют MTP-
FA 51 с GG, аминокислоты 55-522 соответствуют NifB K. oxytoca
согласно работы Temme et al., (2012), а аминокислоты 523-533
<400> 147
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly Met Thr Ser Cys Ser Ser Phe Ser Gly Gly
50 55 60
Lys Ala Cys Arg Pro Ala Asp Asp Ser Ala Leu Thr Pro Leu Val Ala
65 70 75 80
Asp Lys Ala Ala Ala His Pro Cys Tyr Ser Arg His Gly His His Arg
85 90 95
Phe Ala Arg Met His Leu Pro Val Ala Pro Ala Cys Asn Leu Gln Cys
100 105 110
Asn Tyr Cys Asn Arg Lys Phe Asp Cys Ser Asn Glu Ser Arg Pro Gly
115 120 125
Val Ser Ser Thr Leu Leu Thr Pro Glu Gln Ala Val Val Lys Val Arg
130 135 140
Gln Val Ala Gln Ala Ile Pro Gln Leu Ser Val Val Gly Ile Ala Gly
145 150 155 160
Pro Gly Asp Pro Leu Ala Asn Ile Ala Arg Thr Phe Arg Thr Leu Glu
165 170 175
Leu Ile Arg Glu Gln Leu Pro Asp Leu Lys Leu Cys Leu Ser Thr Asn
180 185 190
Gly Leu Met Leu Pro Asp Ala Val Asp Arg Leu Leu Asp Val Gly Val
195 200 205
Asp His Val Thr Val Thr Ile Asn Thr Leu Asp Ala Glu Ile Ala Ala
210 215 220
Gln Ile Tyr Ala Trp Leu Trp Leu Asp Gly Glu Arg Tyr Ser Gly Arg
225 230 235 240
Glu Ala Gly Glu Ile Leu Ile Ala Arg Gln Leu Glu Gly Val Arg Arg
245 250 255
Leu Thr Ala Lys Gly Val Leu Val Lys Ile Asn Ser Val Leu Ile Pro
260 265 270
Gly Ile Asn Asp Ser Gly Met Ala Asp Val Ser Arg Ala Leu Arg Ala
275 280 285
Ser Gly Ala Phe Ile His Asn Ile Met Pro Leu Ile Ala Arg Pro Glu
290 295 300
His Gly Thr Val Phe Gly Leu Asn Gly Gln Pro Glu Pro Asp Ala Glu
305 310 315 320
Thr Leu Ala Ala Thr Arg Ser Arg Cys Gly Glu Val Met Pro Gln Met
325 330 335
Thr His Cys His Gln Cys Arg Ala Asp Ala Ile Gly Met Leu Gly Glu
340 345 350
Asp Arg Ser Gln Gln Phe Thr Gln Leu Pro Ala Pro Glu Ser Leu Pro
355 360 365
Ala Trp Leu Pro Ile Leu His Gln Arg Ala Gln Leu His Ala Ser Ile
370 375 380
Ala Thr Arg Gly Glu Ser Glu Ala Asp Asp Ala Cys Leu Val Ala Val
385 390 395 400
Ala Ser Ser Arg Gly Asp Val Ile Asp Cys His Phe Gly His Ala Asp
405 410 415
Arg Phe Tyr Ile Tyr Ser Leu Ser Ala Ala Gly Met Val Leu Val Asn
420 425 430
Glu Arg Phe Thr Pro Lys Tyr Cys Gln Gly Arg Asp Asp Cys Glu Pro
435 440 445
Gln Asp Asn Ala Ala Arg Phe Ala Ala Ile Leu Glu Leu Leu Ala Asp
450 455 460
Val Lys Ala Val Phe Cys Val Arg Ile Gly His Thr Pro Trp Gln Gln
465 470 475 480
Leu Glu Gln Glu Gly Ile Glu Pro Cys Val Asp Gly Ala Trp Arg Pro
485 490 495
Val Ser Glu Val Leu Pro Ala Trp Trp Gln Gln Arg Arg Gly Ser Trp
500 505 510
Pro Ala Ala Leu Pro His Lys Gly Val Ala Gly Gly Tyr Pro Tyr Asp
515 520 525
Val Pro Asp Tyr Ala
530
<210> 148
<211> 479
<212> PRT
<213> Azospirillum brasilense
<400> 148
Met Ser Leu Ser Val Asn Glu Gly Val Asp Val Lys Gly Leu Val Asp
1. 5 10 15
Lys Val Leu Glu Ala Tyr Pro Glu Lys Ser Arg Lys Arg Arg Ala Lys
20 25 30
His Leu Asn Val Leu Glu Ala Glu Ala Lys Asp Cys Gly Val Lys Ser
35 40 45
Asn Ile Lys Ser Ile Pro Gly Val Met Thr Ile Arg Gly Cys Ala Tyr
50 55 60
Ala Gly Ser Lys Gly Val Val Trp Gly Pro Ile Lys Asp Met Ile His
65 70 75 80
Ile Ser His Gly Pro Val Gly Cys Gly Tyr Tyr Ser Trp Ser Gly Arg
85 90 95
Arg Asn Tyr Tyr Val Gly Asp Thr Gly Val Asp Ser Trp Gly Thr Met
100 105 110
His Phe Thr Ser Asp Phe Gln Glu Lys Asp Ile Val Phe Gly Gly Asp
115 120 125
Lys Lys Leu His Lys Val Ile Glu Glu Ile Asn Glu Leu Phe Pro Leu
130 135 140
Val Asn Gly Ile Ser Ile Gln Ser Glu Cys Pro Ile Gly Leu Ile Gly
145 150 155 160
Asp Asp Ile Glu Ala Val Ala Arg Ala Lys Ser Glu Glu Leu Gly Lys
165 170 175
Pro Val Val Pro Val Arg Cys Glu Gly Phe Arg Gly Val Ser Gln Ser
180 185 190
Leu Gly His His Ile Ala Asn Asp Val Ile Arg Asp Trp Ile Phe Glu
195 200 205
Lys Thr Glu Pro Lys Glu Gly Phe Val Ser Thr Pro Tyr Asp Val Thr
210 215 220
Ile Ile Gly Asp Tyr Asn Ile Gly Gly Asp Ala Trp Ala Ser Arg Ile
225 230 235 240
Leu Leu Glu Glu Ile Gly Leu Arg Val Ile Ala Gln Trp Ser Gly Asp
245 250 255
Gly Thr Leu Ala Glu Leu Glu Asn Thr Pro Lys Ala Lys Val Asn Leu
260 265 270
Ile His Cys Tyr Arg Ser Met Asn Tyr Ile Ala Arg His Met Glu Glu
275 280 285
Lys Phe Gly Ile Pro Trp Met Glu Tyr Asn Phe Phe Gly Pro Ser Gln
290 295 300
Ile Ala Glu Ser Leu Arg Lys Ile Ala Ala Leu Phe Asp Asp Thr Ile
305 310 315 320
Lys Glu Asn Ala Glu Lys Val Ile Ala Lys Tyr Gln Pro Met Val Asp
325 330 335
Ala Val Ile Ala Lys Phe Lys Pro Arg Leu Glu Gly Lys Lys Val Met
340 345 350
Ile Tyr Val Gly Gly Leu Arg Pro Arg His Val Val Asp Ala Tyr His
355 360 365
Asp Leu Gly Met Glu Ile Val Gly Thr Gly Tyr Glu Phe Ala His Asn
370 375 380
Asp Asp Tyr Gln Arg Thr Gln His Tyr Val Lys Glu Gly Thr Leu Ile
385 390 395 400
Tyr Asp Asp Val Thr Ala Phe Glu Leu Glu Lys Phe Val Glu Val Met
405 410 415
Arg Pro Asp Leu Val Ala Ser Gly Ile Lys Glu Lys Tyr Val Phe Gln
420 425 430
Lys Met Gly Leu Pro Phe Arg Gln Met His Ser Trp Asp Tyr Ser Gly
435 440 445
Pro Tyr His Gly Tyr Asp Gly Phe Ala Ile Phe Ala Arg Asp Met Asp
450 455 460
Leu Ala Ile Asn Asn Pro Val Trp Gly Ile Met Lys Ala Pro Phe
465 470 475
<210> 149
<211> 492
<212> PRT
<213> Azotobacter vinelandii
<400> 149
Met Thr Gly Met Ser Arg Glu Glu Val Glu Ser Leu Ile Gln Glu Val
1. 5 10 15
Leu Glu Val Tyr Pro Glu Lys Ala Arg Lys Asp Arg Asn Lys His Leu
20 25 30
Ala Val Asn Asp Pro Ala Val Thr Gln Ser Lys Lys Cys Ile Ile Ser
35 40 45
Asn Lys Lys Ser Gln Pro Gly Leu Met Thr Ile Arg Gly Cys Ala Tyr
50 55 60
Ala Gly Ser Lys Gly Val Val Trp Gly Pro Ile Lys Asp Met Ile His
65 70 75 80
Ile Ser His Gly Pro Val Gly Cys Gly Gln Tyr Ser Arg Ala Gly Arg
85 90 95
Arg Asn Tyr Tyr Ile Gly Thr Thr Gly Val Asn Ala Phe Val Thr Met
100 105 110
Asn Phe Thr Ser Asp Phe Gln Glu Lys Asp Ile Val Phe Gly Gly Asp
115 120 125
Lys Lys Leu Ala Lys Leu Ile Asp Glu Val Glu Thr Leu Phe Pro Leu
130 135 140
Asn Lys Gly Ile Ser Val Gln Ser Glu Cys Pro Ile Gly Leu Ile Gly
145 150 155 160
Asp Asp Ile Glu Ser Val Ser Lys Val Lys Gly Ala Glu Leu Ser Lys
165 170 175
Thr Ile Val Pro Val Arg Cys Glu Gly Phe Arg Gly Val Ser Gln Ser
180 185 190
Leu Gly His His Ile Ala Asn Asp Ala Val Arg Asp Trp Val Leu Gly
195 200 205
Lys Arg Asp Glu Asp Thr Thr Phe Ala Ser Thr Pro Tyr Asp Val Ala
210 215 220
Ile Ile Gly Asp Tyr Asn Ile Gly Gly Asp Ala Trp Ser Ser Arg Ile
225 230 235 240
Leu Leu Glu Glu Met Gly Leu Arg Cys Val Ala Gln Trp Ser Gly Asp
245 250 255
Gly Ser Ile Ser Glu Ile Glu Leu Thr Pro Lys Val Lys Leu Asn Leu
260 265 270
Val His Cys Tyr Arg Ser Met Asn Tyr Ile Ser Arg His Met Glu Glu
275 280 285
Lys Tyr Gly Ile Pro Trp Met Glu Tyr Asn Phe Phe Gly Pro Thr Lys
290 295 300
Thr Ile Glu Ser Leu Arg Ala Ile Ala Ala Lys Phe Asp Glu Ser Ile
305 310 315 320
Gln Lys Lys Cys Glu Glu Val Ile Ala Lys Tyr Lys Pro Glu Trp Glu
325 330 335
Ala Val Val Ala Lys Tyr Arg Pro Arg Leu Glu Gly Lys Arg Val Met
340 345 350
Leu Tyr Ile Gly Gly Leu Arg Pro Arg His Val Ile Gly Ala Tyr Glu
355 360 365
Asp Leu Gly Met Glu Val Val Gly Thr Gly Tyr Glu Phe Ala His Asn
370 375 380
Asp Asp Tyr Asp Arg Thr Met Lys Glu Met Gly Asp Ser Thr Leu Leu
385 390 395 400
Tyr Asp Asp Val Thr Gly Tyr Glu Phe Glu Glu Phe Val Lys Arg Ile
405 410 415
Lys Pro Asp Leu Ile Gly Ser Gly Ile Lys Glu Lys Phe Ile Phe Gln
420 425 430
Lys Met Gly Ile Pro Phe Arg Gln Met His Ser Trp Asp Tyr Ser Gly
435 440 445
Pro Tyr His Gly Phe Asp Gly Phe Ala Ile Phe Ala Arg Asp Met Asp
450 455 460
Met Thr Leu Asn Asn Pro Cys Trp Lys Lys Leu Gln Ala Pro Trp Glu
465 470 475 480
Ala Ser Glu Gly Ala Glu Lys Val Ala Ala Ser Ala
485 490
<210> 150
<211> 504
<212> PRT
<213> Sinorhizobium fredii
<400> 150
Met Ser Leu Asp Tyr Glu Asn Asp Ser Ala Leu His Gln Glu Leu Ile
1. 5 10 15
Thr Gln Val Leu Ser Gln Tyr Pro His Lys Ala Ala Lys Arg Arg Gln
20 25 30
Lys His Leu Ser Val Ala Ser Asp Arg Glu Ala Val Gly Glu Glu Gly
35 40 45
Glu Thr Leu Ser Glu Cys Asp Val Lys Ser Asn Ile Lys Ser Ile Pro
50 55 60
Gly Val Met Thr Ile Arg Gly Cys Ala Tyr Ala Gly Ser Lys Gly Val
65 70 75 80
Val Trp Gly Pro Val Lys Asp Met Val His Ile Ser His Gly Pro Val
85 90 95
Gly Cys Gly Gln Tyr Ser Trp Ser Gln Arg Arg Asn Tyr Tyr Val Gly
100 105 110
Thr Thr Gly Val Asp Thr Phe Val Thr Met Gln Phe Thr Ser Asp Phe
115 120 125
Gln Glu Lys Asp Ile Val Phe Gly Gly Asp Lys Lys Leu Glu Gln Val
130 135 140
Ile Asp Glu Ile Glu Glu Leu Phe Pro Leu Asn Asn Gly Ile Thr Ile
145 150 155 160
Gln Ser Glu Cys Pro Ile Gly Leu Ile Gly Asp Asp Ile Glu Ala Val
165 170 175
Ser Arg Lys Lys Ala Ala Glu His Glu Thr Thr Ile Val Pro Val Arg
180 185 190
Cys Glu Gly Phe Arg Gly Val Ser Gln Ser Leu Gly His His Ile Ala
195 200 205
Asn Asp Ala Ile Arg Asp Trp Val Phe Asp Lys Ala Asp Gly Lys Thr
210 215 220
Asp Val Glu Phe Glu Thr Gly Pro Tyr Asp Val Asn Val Ile Gly Asp
225 230 235 240
Tyr Asn Ile Gly Gly Asp Ala Trp Ala Ser Arg Ile Leu Leu Glu Glu
245 250 255
Ile Gly Leu Arg Val Val Gly Asn Trp Ser Gly Asp Ala Thr Leu Ala
260 265 270
Glu Val Glu Arg Ala Pro Arg Ala Lys Leu Asn Leu Ile His Cys Tyr
275 280 285
Arg Ser Met Asn Tyr Ile Cys Arg His Met Glu Glu Arg Tyr Ala Ile
290 295 300
Pro Trp Met Glu Tyr Asn Phe Phe Gly Pro Ser Gln Ile Glu Ala Ser
305 310 315 320
Leu Arg Lys Ile Ala Arg His Phe Gly Pro Thr Ile Glu Glu Arg Ala
325 330 335
Glu Arg Val Ile Ala Lys Tyr Arg Pro Leu Val Asp Ala Val Ile Asp
340 345 350
Lys Tyr Trp Pro Arg Leu Gln Gly Lys Arg Val Met Leu Tyr Val Gly
355 360 365
Gly Leu Arg Pro Arg His Val Ile Thr Ala Tyr Glu Asp Leu Gly Met
370 375 380
Gln Ile Val Gly Thr Gly Tyr Glu Phe Ala His Asn Asp Asp Tyr Gln
385 390 395 400
Arg Thr Gly His Tyr Val Lys Thr Gly Thr Leu Ile Tyr Asp Asp Ala
405 410 415
Thr Ser Tyr Glu Leu Asp Thr Phe Ile Glu Arg Ile Arg Pro Asp Leu
420 425 430
Val Gly Ser Gly Ile Lys Glu Lys Tyr Pro Val Gln Lys Met Gly Ile
435 440 445
Pro Phe Arg Gln Met His Ser Trp Asp Tyr Ser Gly Pro Tyr His Gly
450 455 460
Tyr Asp Gly Phe Ala Ile Phe Ala Arg Asp Met Asp Leu Ala Ile Asn
465 470 475 480
Asn Pro Val Trp Asp Leu Tyr Asp Ala Pro Trp Lys Lys Met Thr Val
485 490 495
Pro Thr Ala Ala Val Ala Ala Glu
500
<210> 151
<211> 543
<212> PRT
<213> Chlorobium tepidum
<400> 151
Met Glu Ala Lys Val Leu Ile Pro Asp Pro Ser Lys Ile Lys Glu Glu
1. 5 10 15
Leu Ile Asn Lys Tyr Pro Ala Lys Val Ala Lys Lys Arg Ser Lys Ser
20 25 30
Ile Val Val Asn Asp Pro Glu Ile Val Pro Glu Val Gln Ala Asn Val
35 40 45
Arg Thr Val Pro Gly Ile Ile Thr Gln Arg Gly Cys Ala Tyr Ala Gly
50 55 60
Cys Lys Gly Val Val Leu Gly Pro Thr Arg Asp Ile Val Asn Ile Val
65 70 75 80
His Gly Pro Ile Gly Cys Ser Phe Tyr Ala Trp Leu Thr Arg Arg Asn
85 90 95
Gln Thr Arg Pro Glu Thr Pro Glu His Glu Asn Tyr Ile Thr Tyr Cys
100 105 110
Phe Ser Thr Asp Met Gln Glu Glu His Val Val Phe Gly Gly Glu Lys
115 120 125
Lys Leu Lys Val Ala Ile Gln Glu Ala Tyr Asp Leu Phe His Pro Lys
130 135 140
Ala Ile Ala Ile Phe Ser Thr Cys Pro Val Gly Leu Ile Gly Asp Asp
145 150 155 160
Val His Ala Val Ala Arg Glu Met Lys Glu Lys Leu Gly Asp Cys Asn
165 170 175
Val Phe Gly Phe Ser Cys Glu Gly Tyr Arg Gly Val Ser Gln Ser Ala
180 185 190
Gly His His Ile Ala Asn Asn Gly Val Phe Lys His Met Val Gly Asn
195 200 205
Asn Asn Glu Val Lys Pro Gly Lys Phe Lys Leu Asn Leu Leu Gly Glu
210 215 220
Tyr Asn Ile Gly Gly Asp Ala Phe Glu Ile Glu Arg Leu Leu Glu Lys
225 230 235 240
Cys Gly Ile Thr Leu Val Ala Ser Phe Ser Gly Asn Ser Thr Val Gly
245 250 255
Ala Ile Glu Asn Ala His Thr Ala Asp Leu Asn Val Ile Met Cys His
260 265 270
Arg Ser Ile Asn Tyr Met Gly Asp Met Met Glu Thr Lys Tyr Gly Ile
275 280 285
Pro Trp Met Lys Val Asn Phe Val Gly Ala Glu Ser Thr Ala Lys Ser
290 295 300
Leu Arg Lys Ile Ala Glu Tyr Phe Gly Asp Glu Glu Leu Lys Ala Lys
305 310 315 320
Val Glu Glu Val Ile Ala Glu Glu Val Pro Ala Val Lys Ala Ile Ile
325 330 335
Asp Glu Ile Arg Pro Arg Thr Glu Gly Lys Thr Ala Met Leu Phe Val
340 345 350
Gly Gly Ser Arg Ala His His Tyr Gln Asp Leu Phe Ser Glu Leu Gly
355 360 365
Met Thr Thr Ile Ala Ala Gly Tyr Glu Phe Ala His Arg Asp Asp Tyr
370 375 380
Glu Gly Arg Glu Val Leu Pro Lys Ile Lys Ile Asp Ala Asp Ser Lys
385 390 395 400
Asn Ile Glu Glu Leu Lys Val Thr Ala Asp Pro Glu Leu Tyr Asn Pro
405 410 415
Arg Lys Ser Lys Ala Glu Leu Glu Glu Leu Lys Ala Lys Gly Leu Glu
420 425 430
Ile Asn Gly Tyr Glu Gly Met Met Lys Gln Met Met Lys Lys Thr Leu
435 440 445
Val Val Asp Asp Ile Ser His Tyr Glu Ser Glu Lys Leu Ile Glu Met
450 455 460
Tyr Lys Pro Asp Ile Phe Cys Ala Gly Ile Lys Glu Lys Tyr Val Val
465 470 475 480
Gln Lys Met Gly Val Pro Leu Lys Gln Leu His Ser Tyr Asp Tyr Gly
485 490 495
Gly Pro Tyr Thr Gly Phe Lys Gly Ala Val Asn Phe Tyr Lys Asp Ile
500 505 510
Asp Arg Met Val Asn Asn Pro Val Trp Lys Met Ile Lys Ala Pro Trp
515 520 525
Glu Lys Ser Glu Pro Glu Ser Leu Glu Ala Ser Tyr Val Ala Ser
530 535 540
<210> 152
<211> 544
<212> PRT
<213> Desulfovibrio vulgaris
<400> 152
Met Ala Leu Lys His Lys Ser Ile Pro Asp Val Ala Thr Val Lys Glu
1. 5 10 15
Glu Leu Leu Lys Lys Tyr Pro Thr Lys Val Ala Arg Lys Arg Ala Lys
20 25 30
Gln Ile Val Ile Asn Asp Val Lys Asp Gly Asp Val Val Pro Glu Val
35 40 45
Gln Ala Asn Val Arg Thr Thr Pro Gly Ile Ile Thr Met Arg Gly Cys
50 55 60
Thr Tyr Ala Gly Cys Lys Gly Val Ile Leu Gly Pro Thr Arg Asp Ile
65 70 75 80
Val Asn Ile Thr His Gly Pro Ile Gly Cys Gly Phe Tyr Ser Trp Leu
85 90 95
Thr Arg Arg Asn Gln Thr Lys Ala Pro Leu Glu Ser Ser Glu Asn Phe
100 105 110
Met Pro Tyr Ala Phe Ser Thr Asp Met Gln Asp Glu Asp Ile Ile Phe
115 120 125
Gly Gly Glu Lys Lys Leu Ile Ala Ala Ile Gln Glu Ala Tyr Asp Thr
130 135 140
Phe His Pro Lys Ala Ile Ala Ile Phe Ala Thr Cys Pro Val Gly Leu
145 150 155 160
Ile Gly Asp Asp Ile His Ala Val Ala Arg Lys Met Lys Glu Lys Leu
165 170 175
Gly Ile Asn Ile Phe Ala Phe Ser Cys Glu Gly Tyr Lys Gly Val Ser
180 185 190
Gln Ser Ala Gly His His Ile Ala Asn Asn Gln Ile Phe Thr His Val
195 200 205
Val Gly Glu Asp Asp Thr Pro Lys Leu Gly Glu Tyr Lys Ile Asn Met
210 215 220
Leu Gly Glu Tyr Asn Ile Gly Gly Asp Ala Phe Glu Leu Glu Arg Val
225 230 235 240
Leu Glu Lys Cys Gly Ile Thr Leu Val Ser Thr Phe Ser Gly Asn Ser
245 250 255
Thr Tyr Glu His Phe Ala Thr Ala His Gln Ala Asp Leu Asn Ala Val
260 265 270
Met Cys His Arg Ser Ile Asn Tyr Val Ala Glu Met Met Glu Thr Lys
275 280 285
Tyr Gly Ile Pro Trp Ile Lys Val Asn Phe Ile Gly Ala Glu Ser Ser
290 295 300
Ala Lys Ser Leu Arg Lys Ile Ala Gln Tyr Phe Gly Asp Lys Lys Leu
305 310 315 320
Ile Asp Arg Val Glu Glu Val Ile Ala Glu Glu Met Pro Ala Val His
325 330 335
Ala Ala Leu Glu Asp Val Lys Pro Phe Thr Glu Gly Lys Thr Ala Met
340 345 350
Leu Phe Val Gly Gly Ser Arg Ala His His Tyr Gln Asp Leu Phe Thr
355 360 365
Glu Met Gly Met Lys Thr Ile Ala Ala Gly Tyr Glu Phe Ala His Arg
370 375 380
Asp Asp Tyr Glu Gly Arg Lys Val Met Pro Thr Ile Lys Val Asp Ala
385 390 395 400
Asp Ser Arg Asn Ile Glu Glu Ile Glu Val Thr Pro Asp Ser Thr Arg
405 410 415
Phe Val Pro Arg Lys Ser Asp Glu Asp Leu Lys Arg Leu Ala Glu Ala
420 425 430
Gly Phe Thr Phe Lys Asp Tyr Glu Gly Met Met Pro Gln Met Glu Ser
435 440 445
Asp Thr Leu Val Ile Asp Asp Leu Asn Gln Tyr Glu Ala Asp Lys Leu
450 455 460
Ile Glu Leu Leu Lys Pro Asp Val Phe Cys Ala Gly Ile Lys Glu Lys
465 470 475 480
Phe Ser Val Gln Lys Met Gly Val Pro Met Lys Gln Leu His Ser Tyr
485 490 495
Asp Tyr Gly Gly Pro Tyr Ala Gly Phe Lys Gly Ala Val Asn Phe Tyr
500 505 510
Thr Glu Ile Lys Arg Leu Val Thr Ser Lys Val Trp Ser Asp Leu Lys
515 520 525
Ala Pro Trp Glu Glu Asn Pro Glu Leu Ser Ala Thr Tyr Val Trp Glu
530 535 540
<210> 153
<211> 538
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность Desulfotomaculum ferrireducens
дикого типа полипептида NifD, 539aa
<400> 153
Met Ala Ile Asn Glu Lys Val Leu Asp Glu Ile Leu Ser Gln Tyr Pro
1. 5 10 15
Thr Lys Val Lys Lys Asn Arg Lys Lys His Ile Ile Ile Lys Asp Pro
20 25 30
Asn Gln Ala Arg Gln Glu Ile Glu Ala Asn Thr Arg Thr Ile Pro Gly
35 40 45
Ile Ile Thr Asn Arg Gly Cys Ala Tyr Ala Gly Cys Lys Gly Val Val
50 55 60
Leu Gly Pro Leu Lys Asp Val Val His Ile Thr His Gly Pro Ile Gly
65 70 75 80
Cys Gly Tyr Tyr Ser Trp Leu Thr Arg Arg Asn Lys Ala Ala Ser Ser
85 90 95
Asp Pro Thr Lys Asn Phe Ile Ser Tyr Cys Phe Ser Thr Asp Met Gln
100 105 110
Glu Ser Asp Ile Val Phe Gly Gly Glu Lys Lys Leu Ala Arg Met Ile
115 120 125
Asp Glu Val Met Glu Ile Phe Lys Pro Asn Ala Ile Thr Ile Ser Ala
130 135 140
Thr Cys Pro Val Gly Leu Ile Gly Asp Asp Leu Gly Ala Val Ala Lys
145 150 155 160
Ala Ala Glu Gln Lys His Gly Ile Thr Val Met His Phe Asn Cys Glu
165 170 175
Gly Tyr Lys Gly Val Ser Gln Ser Ala Gly His His Ile Ala Asn Asn
180 185 190
Thr Leu Met Glu Arg Val Ile Gly Ala Gly Glu Leu Glu Ala Ala Pro
195 200 205
Gly Arg Tyr Pro Ile Asn Ile Leu Gly Glu Tyr Asn Ile Gly Gly Asp
210 215 220
Ser Trp Glu Ile Glu Arg Ile Leu Arg Glu Ile Gly Tyr Thr Val Leu
225 230 235 240
Ser Val Met Thr Gly Asp Gly Ser Tyr Glu Glu Leu Lys Asn Ala His
245 250 255
Val Ala Glu Leu Asn Leu Val Gln Cys His Arg Ser Ile Asn Tyr Ile
260 265 270
Ala Glu Met Leu Glu Thr Lys Tyr Gly Thr Pro Trp Leu Lys Val Asn
275 280 285
Phe Ile Gly Ile Gln Ser Thr Ile Asp Ser Leu Arg Asn Met Ala Ile
290 295 300
Tyr Phe Gly Asp Pro Glu Leu Thr Arg Arg Thr Glu Glu Val Ile Ala
305 310 315 320
Lys Glu Leu Ala Glu Val Glu Pro Val Met Glu Gln Tyr Lys Lys Ile
325 330 335
Cys Gln Gly Lys Thr Ala Phe Cys Phe Val Gly Gly Ser Arg Gly His
340 345 350
His Tyr Gln Gly Leu Phe Ala Glu Leu Gly Met Glu Thr Val Leu Ala
355 360 365
Gly Tyr Glu Phe Ala His Arg Asp Asp Tyr Glu Gly Arg Asp Val Leu
370 375 380
Pro Gln Ile Lys Leu Asp Ala Asp Asn Lys Asn Ile Pro Glu Leu His
385 390 395 400
Val Glu Pro Asp Gln Arg Arg Phe Lys Leu Lys Val Pro Arg Glu Arg
405 410 415
Met Glu Glu Leu Lys Lys Lys Ile Pro Leu Ser Tyr Tyr Ala Gly Met
420 425 430
Met Val Asp Met Lys Gly Gly His Val Val Val Asp Asp Leu Asn His
435 440 445
Tyr Glu Thr Glu Gln Phe Ile Lys Leu Leu Lys Pro Asp Ile Phe Ala
450 455 460
Ser Gly Ile Lys Asp Lys Tyr Val Val Gln Lys Met Gly Ile Pro Ala
465 470 475 480
Lys Gln Leu His Ser Tyr Asp Tyr Ser Gly Pro Tyr Ala Gly Phe Lys
485 490 495
Gly Ala Val Lys Phe Ala Glu Asp Ile Thr Met Ser Phe Ile Ser Pro
500 505 510
Thr Trp Asn Phe Ile Thr Pro Pro Trp Lys Asn Gln Pro Ile Leu Glu
515 520 525
Gly Glu Ile Val Glu Gly Gly Cys Ser Thr
530 535
<210> 154
<211> 4
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептидная последовательность
<220>
<221> X
<222> (4)..(4)
<223> любая аминокислота, отличная от Y
<400> 154
Arg Arg Asn Xaa
1
<210> 155
<211> 9
<212> PRT
<213> Искусственная последовательность
<220>
<223> Последовательность триптического пептида из NifM
<400> 155
Asp Ala Phe Ala Pro Leu Ala Gln Arg
1. 5
<210> 156
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> Последовательность триптического пептида из NifM
<400> 156
Asp Tyr Leu Trp Gln Gln Ser Gln Gln Arg
1. 5 10
<210> 157
<211> 14
<212> PRT
<213> Искусственная последовательность
<220>
<223> Последовательность триптического пептида из полипептида CAT
<400> 157
Ile Thr Gly Tyr Thr Thr Val Asp Ile Ser Gln Trp His Arg
1. 5 10
<210> 158
<211> 9
<212> PRT
<213> Искусственная последовательность
<220>
<223> Последовательность триптического пептида из полипептида CAT
<400> 158
Leu Met Asn Ala His Pro Glu Phe Arg
1. 5
<210> 159
<211> 7
<212> PRT
<213> Искусственная последовательность
<220>
<223> Последовательность триптического пептида из полипептида CAT
<400> 159
Tyr Tyr Thr Gln Gly Asp Lys
1. 5
<210> 160
<211> 358
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-
FA?51::NifU::TwinStrep, кодированная SN166. Аминокислоты 1-54
представляют собой последовательность MTP-FA?51 с дополнительным
началом трансляции метионина и C-концевым GG, аминокислоты 55-328
представляют собой последовательность NifU, и
<400> 160
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly Met Trp Asn Tyr Ser Glu Lys Val Lys Asp
50 55 60
His Phe Phe Asn Pro Arg Asn Ala Arg Val Val Asp Asn Ala Asn Ala
65 70 75 80
Val Gly Asp Val Gly Ser Leu Ser Cys Gly Asp Ala Leu Arg Leu Met
85 90 95
Leu Arg Val Asp Pro Gln Ser Glu Ile Ile Glu Glu Ala Gly Phe Gln
100 105 110
Thr Phe Gly Cys Gly Ser Ala Ile Ala Ser Ser Ser Ala Leu Thr Glu
115 120 125
Leu Ile Ile Gly His Thr Leu Ala Glu Ala Gly Gln Ile Thr Asn Gln
130 135 140
Gln Ile Ala Asp Tyr Leu Asp Gly Leu Pro Pro Glu Lys Met His Cys
145 150 155 160
Ser Val Met Gly Gln Glu Ala Leu Arg Ala Ala Ile Ala Asn Phe Arg
165 170 175
Gly Glu Ser Leu Glu Glu Glu His Asp Glu Gly Lys Leu Ile Cys Lys
180 185 190
Cys Phe Gly Val Asp Glu Gly His Ile Arg Arg Ala Val Gln Asn Asn
195 200 205
Gly Leu Thr Thr Leu Ala Glu Val Ile Asn Tyr Thr Lys Ala Gly Gly
210 215 220
Gly Cys Thr Ser Cys His Glu Lys Ile Glu Leu Ala Leu Ala Glu Ile
225 230 235 240
Leu Ala Gln Gln Pro Gln Thr Thr Pro Ala Val Ala Ser Gly Lys Asp
245 250 255
Pro His Trp Gln Ser Val Val Asp Thr Ile Ala Glu Leu Arg Pro His
260 265 270
Ile Gln Ala Asp Gly Gly Asp Met Ala Leu Leu Ser Val Thr Asn His
275 280 285
Gln Val Thr Val Ser Leu Ser Gly Ser Cys Ser Gly Cys Met Met Thr
290 295 300
Asp Met Thr Leu Ala Trp Leu Gln Gln Lys Leu Met Glu Arg Thr Gly
305 310 315 320
Cys Tyr Met Glu Val Val Ala Ala Gly Gly Trp Ser His Pro Gln Phe
325 330 335
Glu Lys Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Ser Ala Trp Ser
340 345 350
His Pro Gln Phe Glu Lys
355
<210> 161
<211> 484
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-
FA?51::NifS::TwinStrep, кодированная SN231. Аминокислоты 1-54
представляют собой последовательность MTP-FA?51 с дополнительным
началом трансляции метионина и C-концевым GG, аминокислоты 55-454
представляют собой последовательность NifS, и
<400> 161
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly Met Lys Gln Val Tyr Leu Asp Asn Asn Ala
50 55 60
Thr Thr Arg Leu Asp Pro Met Val Leu Glu Ala Met Met Pro Phe Leu
65 70 75 80
Thr Asp Phe Tyr Gly Asn Pro Ser Ser Ile His Asp Phe Gly Ile Pro
85 90 95
Ala Gln Ala Ala Leu Glu Arg Ala His Gln Gln Ala Ala Ala Leu Leu
100 105 110
Gly Ala Glu Tyr Pro Ser Glu Ile Ile Phe Thr Ser Cys Ala Thr Glu
115 120 125
Ala Thr Ala Thr Ala Ile Ala Ser Ala Ile Ala Leu Leu Pro Glu Arg
130 135 140
Arg Glu Ile Ile Thr Ser Val Val Glu His Pro Ala Thr Leu Ala Ala
145 150 155 160
Cys Glu His Leu Glu Arg Gln Gly Tyr Arg Ile His Arg Ile Ala Val
165 170 175
Asp Ser Glu Gly Ala Leu Asp Met Ala Gln Phe Arg Ala Ala Leu Ser
180 185 190
Pro Arg Val Ala Leu Val Ser Val Met Trp Ala Asn Asn Glu Thr Gly
195 200 205
Val Leu Phe Pro Ile Gly Glu Met Ala Glu Leu Ala His Glu Gln Gly
210 215 220
Ala Leu Phe His Cys Asp Ala Val Gln Val Val Gly Lys Ile Pro Ile
225 230 235 240
Ala Val Gly Gln Thr Arg Ile Asp Met Leu Ser Cys Ser Ala His Lys
245 250 255
Phe His Gly Pro Lys Gly Val Gly Cys Leu Tyr Leu Arg Arg Gly Thr
260 265 270
Arg Phe Arg Pro Leu Leu Arg Gly Gly His Gln Glu Tyr Gly Arg Arg
275 280 285
Ala Gly Thr Glu Asn Ile Cys Gly Ile Val Gly Met Gly Ala Ala Cys
290 295 300
Glu Leu Ala Asn Ile His Leu Pro Gly Met Thr His Ile Gly Gln Leu
305 310 315 320
Arg Asn Arg Leu Glu His Arg Leu Leu Ala Ser Val Pro Ser Val Met
325 330 335
Val Met Gly Gly Gly Gln Pro Arg Val Pro Gly Thr Val Asn Leu Ala
340 345 350
Phe Glu Phe Ile Glu Gly Glu Ala Ile Leu Leu Leu Leu Asn Gln Ala
355 360 365
Gly Ile Ala Ala Ser Ser Gly Ser Ala Cys Thr Ser Gly Ser Leu Glu
370 375 380
Pro Ser His Val Met Arg Ala Met Asn Ile Pro Tyr Thr Ala Ala His
385 390 395 400
Gly Thr Ile Arg Phe Ser Leu Ser Arg Tyr Thr Arg Glu Lys Glu Ile
405 410 415
Asp Tyr Val Val Ala Thr Leu Pro Pro Ile Ile Asp Arg Leu Arg Ala
420 425 430
Leu Ser Pro Tyr Trp Gln Asn Gly Lys Pro Arg Pro Ala Asp Ala Val
435 440 445
Phe Thr Pro Val Tyr Gly Gly Gly Trp Ser His Pro Gln Phe Glu Lys
450 455 460
Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Ser Ala Trp Ser His Pro
465 470 475 480
Gln Phe Glu Lys
<210> 162
<211> 9
<212> PRT
<213> Искусственная последовательность
<220>
<223> Пептидная последовательность
<400> 162
Ile Ser Thr Gln Val Val Arg Asn Met
1. 5
<210> 163
<211> 384
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность полипептида NifV из A. vinelandii
(AvNifV; Учетный №. CP001157); 384aa.
<400> 163
Met Ala Ser Val Ile Ile Asp Asp Thr Thr Leu Arg Asp Gly Glu Gln
1. 5 10 15
Ser Ala Gly Val Ala Phe Asn Ala Asp Glu Lys Ile Ala Ile Ala Arg
20 25 30
Ala Leu Ala Glu Leu Gly Val Pro Glu Leu Glu Ile Gly Ile Pro Ser
35 40 45
Met Gly Glu Glu Glu Arg Glu Val Met His Ala Ile Ala Gly Leu Gly
50 55 60
Leu Ser Ser Arg Leu Leu Ala Trp Cys Arg Leu Cys Asp Val Asp Leu
65 70 75 80
Ala Ala Ala Arg Ser Thr Gly Val Thr Met Val Asp Leu Ser Leu Pro
85 90 95
Val Ser Asp Leu Met Leu His His Lys Leu Asn Arg Asp Arg Asp Trp
100 105 110
Ala Leu Arg Glu Val Ala Arg Leu Val Gly Glu Ala Arg Met Ala Gly
115 120 125
Leu Glu Val Cys Leu Gly Cys Glu Asp Ala Ser Arg Ala Asp Leu Glu
130 135 140
Phe Val Val Gln Val Gly Glu Val Ala Gln Ala Ala Gly Ala Arg Arg
145 150 155 160
Leu Arg Phe Ala Asp Thr Val Gly Val Met Glu Pro Phe Gly Met Leu
165 170 175
Asp Arg Phe Arg Phe Leu Ser Arg Arg Leu Asp Met Glu Leu Glu Val
180 185 190
His Ala His Asp Asp Phe Gly Leu Ala Thr Ala Asn Thr Leu Ala Ala
195 200 205
Val Met Gly Gly Ala Thr His Ile Asn Thr Thr Val Asn Gly Leu Gly
210 215 220
Glu Arg Ala Gly Asn Ala Ala Leu Glu Glu Cys Val Leu Ala Leu Lys
225 230 235 240
Asn Leu His Gly Ile Asp Thr Gly Ile Asp Thr Arg Gly Ile Pro Ala
245 250 255
Ile Ser Ala Leu Val Glu Arg Ala Ser Gly Arg Gln Val Ala Trp Gln
260 265 270
Lys Ser Val Val Gly Ala Gly Val Phe Thr His Glu Ala Gly Ile His
275 280 285
Val Asp Gly Leu Leu Lys His Arg Arg Asn Tyr Glu Gly Leu Asn Pro
290 295 300
Asp Glu Leu Gly Arg Ser His Ser Leu Val Leu Gly Lys His Ser Gly
305 310 315 320
Ala His Met Val Arg Asn Thr Tyr Arg Asp Leu Gly Ile Glu Leu Ala
325 330 335
Asp Trp Gln Ser Gln Ala Leu Leu Gly Arg Ile Arg Ala Phe Ser Thr
340 345 350
Arg Thr Lys Arg Ser Pro Gln Pro Ala Glu Leu Gln Asp Phe Tyr Arg
355 360 365
Gln Leu Cys Glu Gln Gly Asn Pro Glu Leu Ala Ala Gly Gly Met Ala
370 375 380
<210> 164
<211> 380
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность вариантной последовательности KoNifV
<400> 164
Met Glu Arg Val Leu Ile Asn Asp Thr Thr Leu Arg Asp Gly Glu Gln
1. 5 10 15
Ser Pro Gly Val Ala Phe Arg Thr Ser Glu Lys Val Ala Ile Ala Glu
20 25 30
Ala Leu Tyr Ala Ala Gly Ile Thr Ala Met Glu Val Gly Thr Pro Ala
35 40 45
Met Gly Asp Glu Glu Ile Ala Arg Ile Gln Leu Val Arg Arg Gln Leu
50 55 60
Pro Asp Ala Thr Leu Met Thr Trp Cys Arg Met Asn Ala Leu Glu Ile
65 70 75 80
Arg Gln Ser Ala Asp Leu Gly Ile Asp Trp Val Asp Ile Ser Ile Pro
85 90 95
Ala Ser Asp Lys Leu Arg Gln Tyr Lys Leu Arg Glu Pro Leu Ala Val
100 105 110
Leu Leu Glu Arg Leu Ala Met Phe Ile His Leu Ala His Thr Leu Gly
115 120 125
Leu Lys Val Cys Ile Gly Cys Glu Asp Ala Ser Arg Ala Ser Gly Gln
130 135 140
Thr Leu Arg Ala Ile Ala Glu Val Ala Gln Gln Cys Ala Ala Ala Arg
145 150 155 160
Leu Arg Tyr Ala Asp Thr Val Gly Leu Leu Asp Pro Phe Thr Thr Ala
165 170 175
Ala Gln Ile Ser Ala Leu Arg Asp Val Trp Ser Gly Glu Ile Glu Met
180 185 190
His Ala His Asn Asp Leu Gly Met Ala Thr Ala Asn Thr Leu Ala Ala
195 200 205
Val Ser Ala Gly Ala Thr Ser Val Asn Thr Thr Val Leu Gly Leu Gly
210 215 220
Glu Arg Ala Gly Asn Ala Ala Leu Glu Thr Val Ala Leu Gly Leu Glu
225 230 235 240
Arg Cys Leu Gly Val Glu Thr Gly Val His Phe Ser Ala Leu Pro Ala
245 250 255
Leu Cys Gln Arg Val Ala Glu Ala Ala Gln Arg Ala Ile Asp Pro Gln
260 265 270
Gln Pro Leu Val Gly Glu Leu Val Phe Thr His Glu Ser Gly Val His
275 280 285
Val Ala Ala Leu Leu Arg Asp Ser Glu Ser Tyr Gln Ser Ile Ala Pro
290 295 300
Ser Leu Met Gly Arg Ser Tyr Arg Leu Val Leu Gly Lys His Ser Gly
305 310 315 320
Arg Gln Ala Val Asn Gly Val Phe Asp Gln Met Gly Tyr His Leu Asn
325 330 335
Ala Ala Gln Ile Asn Gln Leu Leu Pro Ala Ile Arg Arg Phe Ala Glu
340 345 350
Asn Trp Lys Arg Ser Pro Lys Asp Tyr Glu Leu Val Ala Ile Tyr Asp
355 360 365
Glu Leu Cys Gly Glu Ser Ala Leu Arg Ala Arg Gly
370 375 380
<210> 165
<211> 12
<212> PRT
<213> Искусственная последовательность
<220>
<223> N-концевое продолжение (последовательность scar).
<400> 165
Met Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Gly Gly
1. 5 10
<210> 166
<211> 22
<212> PRT
<213> Искусственная последовательность
<220>
<223> N-концевое продолжение AvNifV (последовательность scar)
<400> 166
Ile Ser Thr Gln Val Val Arg Asn Arg Gly Gly Tyr Pro Tyr Asp Val
1. 5 10 15
Pro Asp Tyr Ala Gly Gly
20
<210> 167
<211> 330
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-
FA?51::HA::KoNifM, кодированная SN43. Аминокислоты 1-53 соответствуют
последовательности MTP-FA?51, включающей GG на ее C-конце,
аминокислоты 54-64 соответствуют эпитопу HA, включающему GG на своем
C-конце,
<400> 167
Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro Ser
1. 5 10 15
Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser Asp
20 25 30
Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val Val
35 40 45
Arg Asn Arg Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Gly Gly
50 55 60
Met Asn Pro Trp Gln Arg Phe Ala Arg Gln Arg Leu Ala Arg Ser Arg
65 70 75 80
Trp Asn Arg Asp Pro Ala Ala Leu Asp Pro Ala Asp Thr Pro Ala Phe
85 90 95
Glu Gln Ala Trp Gln Arg Gln Cys His Met Glu Gln Thr Ile Val Ala
100 105 110
Arg Val Pro Glu Gly Asp Ile Pro Ala Ala Leu Leu Glu Asn Ile Ala
115 120 125
Ala Ser Leu Ala Ile Trp Leu Asp Glu Gly Asp Phe Ala Pro Pro Glu
130 135 140
Arg Ala Ala Ile Val Arg His His Ala Arg Leu Glu Leu Ala Phe Ala
145 150 155 160
Asp Ile Ala Arg Gln Ala Pro Gln Pro Asp Leu Ser Thr Val Gln Ala
165 170 175
Trp Tyr Leu Arg His Gln Thr Gln Phe Met Arg Pro Glu Gln Arg Leu
180 185 190
Thr Arg His Leu Leu Leu Thr Val Asp Asn Asp Arg Glu Ala Val His
195 200 205
Gln Arg Ile Leu Gly Leu Tyr Arg Gln Ile Asn Ala Ser Arg Asp Ala
210 215 220
Phe Ala Pro Leu Ala Gln Arg His Ser His Cys Pro Ser Ala Leu Glu
225 230 235 240
Glu Gly Arg Leu Gly Trp Ile Ser Arg Gly Leu Leu Tyr Pro Gln Leu
245 250 255
Glu Thr Ala Leu Phe Ser Leu Ala Glu Asn Ala Leu Ser Leu Pro Ile
260 265 270
Ala Ser Glu Leu Gly Trp His Leu Leu Trp Cys Glu Ala Ile Arg Pro
275 280 285
Ala Ala Pro Met Glu Pro Gln Gln Ala Leu Glu Ser Ala Arg Asp Tyr
290 295 300
Leu Trp Gln Gln Ser Gln Gln Arg His Gln Arg Gln Trp Leu Glu Gln
305 310 315 320
Met Ile Ser Arg Gln Pro Gly Leu Cys Gly
325 330
<210> 168
<211> 354
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-
CoxIV::TwinStrep::NifH, кодированная SN178. Аминокислоты 1-31
соответствуют последовательности MTP-CoxIV, аминокислоты 32-61
соответствуют последовательности TwinStrep включающей GG на ее C-
конце, и аминокислоты 62-354
<400> 168
Met Leu Ser Leu Arg Gln Ser Ile Arg Phe Phe Lys Pro Ala Thr Arg
1. 5 10 15
Thr Leu Cys Ser Ser Arg Tyr Leu Leu Gln Gln Lys Pro Ser Ala Trp
20 25 30
Ser His Pro Gln Phe Glu Lys Gly Gly Gly Ser Gly Gly Gly Ser Gly
35 40 45
Gly Ser Ala Trp Ser His Pro Gln Phe Glu Lys Gly Gly Met Ser Leu
50 55 60
Arg Gln Ile Ala Phe Tyr Gly Lys Gly Gly Ile Gly Lys Ser Thr Thr
65 70 75 80
Ser Gln Asn Thr Leu Ala Ala Leu Val Glu Leu Asp Gln Lys Ile Leu
85 90 95
Ile Val Gly Cys Asp Pro Lys Ala Asp Ser Thr Arg Leu Ile Leu His
100 105 110
Ala Lys Ala Gln Asp Thr Val Leu His Leu Ala Ala Glu Ala Gly Ser
115 120 125
Val Glu Asp Leu Glu Leu Glu Asp Val Leu Lys Ile Gly Tyr Lys Gly
130 135 140
Ile Lys Cys Val Glu Ser Gly Gly Pro Glu Pro Gly Val Gly Cys Ala
145 150 155 160
Gly Arg Gly Val Ile Thr Ser Ile Asn Phe Leu Glu Glu Asn Gly Ala
165 170 175
Tyr Asp Asp Val Asp Tyr Val Ser Tyr Asp Val Leu Gly Asp Val Val
180 185 190
Cys Gly Gly Phe Ala Met Pro Ile Arg Glu Asn Lys Ala Gln Glu Ile
195 200 205
Tyr Ile Val Met Ser Gly Glu Met Met Ala Leu Tyr Ala Ala Asn Asn
210 215 220
Ile Ala Lys Gly Ile Leu Lys Tyr Ala His Ser Gly Gly Val Arg Leu
225 230 235 240
Gly Gly Leu Ile Cys Asn Glu Arg Gln Thr Asp Lys Glu Ile Asp Leu
245 250 255
Ala Ser Ala Leu Ala Ala Arg Leu Gly Thr Gln Leu Ile His Phe Val
260 265 270
Pro Arg Asp Asn Ile Val Gln His Ala Glu Leu Arg Arg Met Thr Val
275 280 285
Ile Glu Tyr Ala Pro Asp Ser Gln Gln Ala Gln Glu Tyr Arg Gln Leu
290 295 300
Ala Asn Lys Val His Ala Asn Lys Gly Lys Gly Thr Ile Pro Thr Pro
305 310 315 320
Ile Thr Met Glu Glu Leu Glu Glu Met Leu Met Asp Phe Gly Ile Met
325 330 335
Lys Ser Glu Glu Gln Gln Leu Ala Glu Leu Gln Ala Lys Glu Ala Ala
340 345 350
Lys Ala
<210> 169
<211> 356
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-
CoxIV::TwinStrep::NifH, кодированная SN179. Аминокислоты 1-31
соответствуют последовательности MTP-CoxIV, аминокислоты 32-61
соответствуют последовательности TwinStrep включающей GG на ее C-
конце, и аминокислоты 62-356
<400> 169
Met Leu Ser Leu Arg Gln Ser Ile Arg Phe Phe Lys Pro Ala Thr Arg
1. 5 10 15
Thr Leu Cys Ser Ser Arg Tyr Leu Leu Gln Gln Lys Pro Ser Ala Trp
20 25 30
Ser His Pro Gln Phe Glu Lys Gly Gly Gly Ser Gly Gly Gly Ser Gly
35 40 45
Gly Ser Ala Trp Ser His Pro Gln Phe Glu Lys Gly Gly Met Thr Glu
50 55 60
Asn Ile Arg Gln Ile Ala Phe Tyr Gly Lys Gly Gly Ile Gly Lys Ser
65 70 75 80
Thr Thr Ser Gln Asn Thr Leu Ala Ala Met Ala Glu Met Gly Gln Arg
85 90 95
Ile Met Ile Val Gly Cys Asp Pro Lys Ala Asp Ser Thr Arg Leu Met
100 105 110
Leu His Ser Lys Ala Gln Thr Thr Val Leu His Leu Ala Ala Glu Arg
115 120 125
Gly Ala Val Glu Asp Leu Glu Leu Glu Glu Val Met Leu Thr Gly Phe
130 135 140
Arg Gly Val Lys Cys Val Glu Ser Gly Gly Pro Glu Pro Gly Val Gly
145 150 155 160
Cys Ala Gly Arg Gly Ile Ile Thr Ala Ile Asn Phe Leu Glu Glu Asn
165 170 175
Gly Ala Tyr Gln Asp Leu Asp Phe Val Ser Tyr Asp Val Leu Gly Asp
180 185 190
Val Val Cys Gly Gly Phe Ala Met Pro Ile Arg Glu Gly Lys Ala Gln
195 200 205
Glu Ile Tyr Ile Val Thr Ser Gly Glu Met Met Ala Met Tyr Ala Ala
210 215 220
Asn Asn Ile Ala Arg Gly Ile Leu Lys Tyr Ala His Ser Gly Gly Val
225 230 235 240
Arg Leu Gly Gly Leu Ile Cys Asn Ser Arg Lys Val Asp Arg Glu Ala
245 250 255
Glu Leu Ile Glu Asn Leu Ala Glu Arg Leu Asn Thr Gln Met Ile His
260 265 270
Phe Val Pro Arg Asp Asn Ile Val Gln His Ala Glu Leu Arg Arg Met
275 280 285
Thr Val Asn Glu Tyr Ala Pro Asp Ser Asn Gln Ser Gln Glu Tyr Arg
290 295 300
Ala Leu Ala Lys Lys Ile Ile Asn Asn Thr Lys Leu Thr Ile Pro Thr
305 310 315 320
Pro Met Glu Met Asp Glu Leu Glu Ala Leu Leu Ile Glu Tyr Gly Ile
325 330 335
Leu Asp Asp Asp Thr Lys His Ala Asp Ile Ile Gly Lys Pro Ala Glu
340 345 350
Ala Ser Ala Lys
355
<210> 170
<211> 348
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-
CoxIV::TwinStrep::NifH, кодированная SN180. Аминокислоты 1-31
соответствуют последовательности MTP-CoxIV, аминокислоты 32-61
соответствуют последовательности TwinStrep включающей GG на ее C-
конце, и аминокислоты 62-338
<400> 170
Met Leu Ser Leu Arg Gln Ser Ile Arg Phe Phe Lys Pro Ala Thr Arg
1. 5 10 15
Thr Leu Cys Ser Ser Arg Tyr Leu Leu Gln Gln Lys Pro Ser Ala Trp
20 25 30
Ser His Pro Gln Phe Glu Lys Gly Gly Gly Ser Gly Gly Gly Ser Gly
35 40 45
Gly Ser Ala Trp Ser His Pro Gln Phe Glu Lys Gly Gly Met Arg Gln
50 55 60
Ile Ala Phe Tyr Gly Lys Gly Gly Ile Gly Lys Ser Thr Thr Gln Gln
65 70 75 80
Asn Thr Met Ala Ala Met Ala Glu Met Gly Lys Lys Val Met Ile Val
85 90 95
Gly Cys Asp Pro Lys Ala Asp Ser Thr Arg Leu Ile Leu His Ser Lys
100 105 110
Ala Gln Thr Ser Val Ile Gln Leu Ala Ala Glu Lys Gly Ser Val Glu
115 120 125
Asp Leu Glu Leu Asp Glu Val Leu Val Glu Gly Gln Trp Gly Ile Lys
130 135 140
Cys Val Glu Ser Gly Gly Pro Glu Pro Gly Val Gly Cys Ala Gly Arg
145 150 155 160
Gly Val Ile Thr Ser Ile Ser Tyr Leu Glu Glu Ala Gly Ala Tyr Glu
165 170 175
Asp Leu Asp Phe Val Thr Tyr Asp Val Leu Gly Asp Val Val Cys Gly
180 185 190
Gly Phe Ala Met Pro Ile Arg Gln Gly Lys Ala Gln Glu Ile Tyr Ile
195 200 205
Val Thr Ser Gly Glu Met Met Ala Met Tyr Ala Ala Asn Asn Ile Ala
210 215 220
Arg Gly Ile Leu Lys Tyr Ala His Ser Gly Gly Val Arg Leu Gly Gly
225 230 235 240
Leu Ile Cys Asn Ser Arg Asn Thr Asp Arg Glu Asp Glu Leu Ile Ile
245 250 255
Glu Leu Ala Arg Arg Leu Asn Thr Gln Met Ile His Phe Ile Pro Arg
260 265 270
Asn Asn Val Val Gln His Ala Glu Leu Arg Arg Met Thr Val Ile Glu
275 280 285
Tyr Asp Pro Lys Asn Glu Gln Ala Asp Gln Tyr Arg Gln Leu Ala Lys
290 295 300
Lys Ile Val Asp Asn Asp Met Lys Thr Ile Pro Thr Pro Ile Thr Met
305 310 315 320
Asp Glu Leu Glu Glu Leu Leu Ile Glu Phe Gly Ile Met Glu Gln Glu
325 330 335
Asp Glu Ser Ile Ile Gly Lys Ala Ala Ala Val Ala
340 345
<210> 171
<211> 354
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-
CoxIV::TwinStrep::NifH, кодированная SN181. Аминокислоты 1-31
соответствуют последовательности MTP-CoxIV, аминокислоты 32-61
соответствуют последовательности TwinStrep включающей GG на ее C-
конце, и аминокислоты 62-338
<400> 171
Met Leu Ser Leu Arg Gln Ser Ile Arg Phe Phe Lys Pro Ala Thr Arg
1. 5 10 15
Thr Leu Cys Ser Ser Arg Tyr Leu Leu Gln Gln Lys Pro Ser Ala Trp
20 25 30
Ser His Pro Gln Phe Glu Lys Gly Gly Gly Ser Gly Gly Gly Ser Gly
35 40 45
Gly Ser Ala Trp Ser His Pro Gln Phe Glu Lys Gly Gly Met Ala Thr
50 55 60
Arg Gln Cys Ala Ile Tyr Gly Lys Gly Gly Ile Gly Lys Ser Thr Thr
65 70 75 80
Thr Gln Asn Leu Val Ala Gly Leu Ala Glu Leu Gly Lys Arg Val Met
85 90 95
Ile Val Gly Cys Asp Pro Lys Ala Asp Ser Thr Arg Leu Ile Leu His
100 105 110
Ser Lys Ala Gln Glu Thr Ile Met Gln Met Ala Ala Asp Ala Gly Ser
115 120 125
Val Glu Asp Leu Glu Leu Glu Asp Val Leu Lys Val Gly Phe Gly Asp
130 135 140
Ile Lys Cys Val Glu Ser Gly Gly Pro Glu Pro Gly Val Gly Cys Ala
145 150 155 160
Gly Arg Gly Val Ile Thr Ala Ile Asn Phe Leu Glu Glu Glu Gly Ala
165 170 175
Tyr Glu Glu Asp Leu Asp Phe Val Phe Tyr Asp Val Leu Gly Asp Val
180 185 190
Val Cys Gly Gly Phe Ala Met Pro Ile Arg Glu Asn Lys Ala Gln Glu
195 200 205
Ile Tyr Ile Val Cys Ser Gly Glu Met Met Ala Met Tyr Ala Ala Asn
210 215 220
Asn Ile Ala Lys Gly Ile Val Lys Tyr Ala Ser Ser Gly Gly Val Arg
225 230 235 240
Leu Ala Gly Leu Ile Cys Asn Ser Arg Asn Thr Ala Arg Glu Asp Glu
245 250 255
Leu Ile Met Glu Leu Ala Arg Gln Leu Gly Thr Gln Met Ile His Phe
260 265 270
Val Pro Arg Asp Asn Ile Val Gln Arg Ala Glu Ile Arg Arg Met Thr
275 280 285
Val Ile Glu Tyr Asp Pro Lys Ser Gly Gln Ala Asp Glu Tyr Arg Thr
290 295 300
Leu Ala Gln Lys Ile Ile Asp Asn Lys Met Phe Val Val Pro Thr Pro
305 310 315 320
Ile Ser Met Asp Ala Leu Glu Asp Leu Leu Met Glu Phe Gly Val Leu
325 330 335
Glu Glu Glu Asp Glu Ser Ile Val Gly Lys Thr Ala Ala Glu Glu Ala
340 345 350
Val Ala
<210> 172
<211> 345
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-
CoxIV::TwinStrep::NifH, кодированная SN182. Аминокислоты 1-31
соответствуют последовательности MTP-CoxIV, аминокислоты 32-61
соответствуют последовательности TwinStrep включающей GG на ее C-
конце, и аминокислоты 62-345
<400> 172
Met Leu Ser Leu Arg Gln Ser Ile Arg Phe Phe Lys Pro Ala Thr Arg
1. 5 10 15
Thr Leu Cys Ser Ser Arg Tyr Leu Leu Gln Gln Lys Pro Ser Ala Trp
20 25 30
Ser His Pro Gln Phe Glu Lys Gly Gly Gly Ser Gly Gly Gly Ser Gly
35 40 45
Gly Ser Ala Trp Ser His Pro Gln Phe Glu Lys Gly Gly Met Ser Phe
50 55 60
Asp Glu Ile Ala Pro Asn Ala Lys Lys Val Ala Ile Tyr Gly Lys Gly
65 70 75 80
Gly Ile Gly Lys Ser Thr Thr Thr Gln Asn Thr Ala Ala Ala Leu Ala
85 90 95
Tyr Tyr Tyr Lys Leu Lys Gly Met Ile His Gly Cys Asp Pro Lys Ala
100 105 110
Asp Ser Thr Arg Met Ile Leu His Gly Lys Pro Gln Glu Thr Val Met
115 120 125
Asp Val Leu Arg Glu Glu Gly Glu Glu Gly Val Thr Leu Glu Lys Val
130 135 140
Arg Lys Val Gly Phe Cys Gly Ile Tyr Cys Val Glu Ser Gly Gly Pro
145 150 155 160
Glu Pro Gly Val Gly Cys Ala Gly Arg Gly Val Ile Thr Ala Val Asn
165 170 175
Leu Met Lys Glu Leu Gly Gly Tyr Pro Asp Asp Leu Asp Phe Leu Phe
180 185 190
Phe Asp Val Leu Gly Asp Val Val Cys Gly Gly Phe Ala Met Pro Leu
195 200 205
Arg Asp Gly Leu Ala Lys Glu Ile Tyr Ile Val Ser Ser Gly Glu Met
210 215 220
Met Ala Leu Tyr Ala Ala Asn Asn Ile Ala Lys Gly Ile Leu Lys Tyr
225 230 235 240
Ala Glu Gln Ser Gly Val Arg Leu Gly Gly Ile Ile Cys Asn Ser Arg
245 250 255
Asn Val Asp Gly Glu Arg Glu Leu Met Glu Glu Phe Cys Asp Lys Leu
260 265 270
Gly Thr Lys Leu Ile His Phe Ile Pro Arg Asp Asn Ile Val Gln Lys
275 280 285
Ala Glu Phe Asn Lys Met Thr Val Val Glu Phe Ala Pro Asp His Pro
290 295 300
Gln Ala Leu Glu Tyr Lys Lys Leu Gly Lys Lys Ile Met Asp Asn Asp
305 310 315 320
Glu Leu Val Ile Pro Thr Pro Leu Ser Met Asp Glu Leu Glu Lys Leu
325 330 335
Val Glu Lys Tyr Gly Leu Tyr Asp Lys
340 345
<210> 173
<211> 345
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-
CoxIV::TwinStrep::NifH, кодированная SN183. Аминокислоты 1-31
соответствуют последовательности MTP-CoxIV, аминокислоты 32-61
соответствуют последовательности TwinStrep включающей GG на ее C-
конце, и аминокислоты 62-345
<400> 173
Met Leu Ser Leu Arg Gln Ser Ile Arg Phe Phe Lys Pro Ala Thr Arg
1. 5 10 15
Thr Leu Cys Ser Ser Arg Tyr Leu Leu Gln Gln Lys Pro Ser Ala Trp
20 25 30
Ser His Pro Gln Phe Glu Lys Gly Gly Gly Ser Gly Gly Gly Ser Gly
35 40 45
Gly Ser Ala Trp Ser His Pro Gln Phe Glu Lys Gly Gly Met Arg Gln
50 55 60
Ile Ala Ile Tyr Gly Lys Gly Gly Ile Gly Lys Ser Thr Thr Thr Gln
65 70 75 80
Asn Thr Val Ser Ala Leu Ala Glu Met Gly Lys Lys Val Met Ile Val
85 90 95
Gly Cys Asp Pro Lys Ala Asp Ser Thr Arg Leu Ile Leu His Ser Lys
100 105 110
Ala Gln Ala Thr Val Met Asp Leu Ala Arg Glu Lys Gly Thr Val Glu
115 120 125
Asp Leu Glu Leu Ser Asp Val Leu Leu Thr Gly Phe Ala Asp Ile Arg
130 135 140
Cys Ala Glu Ser Gly Gly Pro Glu Pro Gly Val Gly Cys Ala Gly Arg
145 150 155 160
Gly Val Ile Thr Ala Ile Asn Phe Leu Glu Glu Asn Gly Ala Tyr Thr
165 170 175
Pro Asp Leu Asp Tyr Val Phe Tyr Asp Val Leu Gly Asp Val Val Cys
180 185 190
Gly Gly Phe Ala Met Pro Ile Arg Glu Asn Lys Ala Gln Glu Ile Tyr
195 200 205
Ile Val Thr Ser Gly Glu Met Met Ala Met Tyr Ala Ala Asn Asn Ile
210 215 220
Ala Arg Gly Ile Leu Lys Tyr Ala Ser Ser Gly Lys Val Arg Leu Gly
225 230 235 240
Gly Leu Ile Cys Asn Ser Arg Lys Val Asp Lys Glu Tyr Glu Leu Ile
245 250 255
Asp Glu Leu Ala Thr Arg Leu Gly Thr Gln Met Ile His Phe Leu Pro
260 265 270
Arg Asp Asn Gln Val Gln Arg Ala Glu Leu Arg Arg Met Thr Val Ile
275 280 285
Glu Tyr Ser Pro Asp His Pro Gln Ala Asp Glu Tyr Arg Ala Leu Ala
290 295 300
Lys Lys Ile Asp Glu Asn Lys Lys Leu Val Ile Pro Thr Pro Leu Thr
305 310 315 320
Met Asp Glu Leu Glu Asp Leu Leu Ile Gln Tyr Gly Ile Leu Glu Asp
325 330 335
Glu Glu Thr Ala Ala Ala Lys Leu Gly
340 345
<210> 174
<211> 335
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-
CoxIV::TwinStrep::NifH, кодированная SN184. Аминокислоты 1-31
соответствуют последовательности MTP-CoxIV, аминокислоты 32-61
соответствуют последовательности TwinStrep включающей GG на ее C-
конце, и аминокислоты 62-335
<400> 174
Met Leu Ser Leu Arg Gln Ser Ile Arg Phe Phe Lys Pro Ala Thr Arg
1. 5 10 15
Thr Leu Cys Ser Ser Arg Tyr Leu Leu Gln Gln Lys Pro Ser Ala Trp
20 25 30
Ser His Pro Gln Phe Glu Lys Gly Gly Gly Ser Gly Gly Gly Ser Gly
35 40 45
Gly Ser Ala Trp Ser His Pro Gln Phe Glu Lys Gly Gly Met Arg Lys
50 55 60
Val Ala Ile Tyr Gly Lys Gly Gly Ile Gly Lys Ser Thr Thr Thr Gln
65 70 75 80
Asn Thr Val Ala Gly Leu Ala Glu Ala Gly Lys Lys Val Met Val Val
85 90 95
Gly Cys Asp Pro Lys Ala Asp Ser Thr Arg Leu Leu Leu Gly Gly Leu
100 105 110
Gln Gln Lys Thr Val Leu Asp Thr Leu Arg Glu Glu Gly Glu Glu Val
115 120 125
Glu Leu Glu Asp Ile Ile Lys Glu Gly Tyr Arg Asn Thr Arg Cys Thr
130 135 140
Glu Ser Gly Gly Pro Glu Pro Gly Val Gly Cys Ala Gly Arg Gly Ile
145 150 155 160
Ile Thr Ser Val Asn Leu Leu Glu Gln Leu Gly Ala Tyr Asp Asp Glu
165 170 175
Trp Glu Leu Asp Tyr Val Phe Tyr Asp Val Leu Gly Asp Val Val Cys
180 185 190
Gly Gly Phe Ala Met Pro Ile Arg Asp Gly Lys Ala Glu Glu Ile Tyr
195 200 205
Ile Val Cys Ser Gly Glu Met Met Ala Met Tyr Ala Ala Asn Asn Ile
210 215 220
Cys Lys Gly Ile Leu Lys Tyr Ala Asp Ala Gly Gly Val Arg Leu Gly
225 230 235 240
Gly Leu Ile Cys Asn Ser Arg Lys Val Asp Asn Glu Arg Glu Met Ile
245 250 255
Glu Glu Leu Ala Arg Arg Leu Gly Thr Gln Met Ile His Phe Val Pro
260 265 270
Arg Asp Asn Phe Val Gln Arg Ala Glu Ile Asn Arg Lys Thr Val Ile
275 280 285
Asp Phe Asp Pro Thr His Pro Gln Ala Asp Glu Tyr Arg Ala Leu Ala
290 295 300
Lys Lys Ile Asp Glu Asn Lys Met Phe Val Ile Pro Lys Pro Leu Glu
305 310 315 320
Ile Asp Glu Leu Glu Ser Leu Leu Ile Glu Phe Gly Ile Ala Asn
325 330 335
<210> 175
<211> 350
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-
CoxIV::TwinStrep::NifH, кодированная SN185. Аминокислоты 1-31
соответствуют последовательности MTP-CoxIV, аминокислоты 32-61
соответствуют последовательности TwinStrep включающей GG на ее
C-конце, и аминокислоты 62-350
<400> 175
Met Leu Ser Leu Arg Gln Ser Ile Arg Phe Phe Lys Pro Ala Thr Arg
1. 5 10 15
Thr Leu Cys Ser Ser Arg Tyr Leu Leu Gln Gln Lys Pro Ser Ala Trp
20 25 30
Ser His Pro Gln Phe Glu Lys Gly Gly Gly Ser Gly Gly Gly Ser Gly
35 40 45
Gly Ser Ala Trp Ser His Pro Gln Phe Glu Lys Gly Gly Met Arg Gln
50 55 60
Ile Ala Ile Tyr Gly Lys Gly Gly Ile Gly Lys Ser Thr Thr Thr Gln
65 70 75 80
Asn Thr Val Ala Gly Leu Ala Ser Ile Gly Lys Lys Val Met Ile Val
85 90 95
Gly Cys Asp Pro Lys Ala Asp Ser Thr Arg Leu Ile Leu His Ala Lys
100 105 110
Ala Gln Ser Thr Val Met Asp Leu Val Arg Glu Leu Gly Thr Val Glu
115 120 125
Asp Leu Glu Leu Glu Asp Val Met Lys Val Gly Tyr Gly Asp Val Lys
130 135 140
Cys Val Glu Ser Gly Gly Pro Glu Pro Gly Val Gly Cys Ala Gly Arg
145 150 155 160
Gly Val Ile Thr Ala Ile Asn Phe Leu Glu Glu Asn Gly Ala Tyr Thr
165 170 175
Pro Asp Leu Asp Phe Val Phe Tyr Asp Val Leu Gly Asp Val Val Cys
180 185 190
Gly Gly Phe Ala Met Pro Ile Arg Glu Gly Lys Ala Glu Glu Ile Tyr
195 200 205
Ile Val Cys Ser Gly Glu Met Met Ala Met Tyr Ala Ala Asn Asn Ile
210 215 220
Ala Lys Gly Ile Leu Lys Tyr Ala Thr Ser Gly Lys Val Arg Leu Ala
225 230 235 240
Gly Leu Ile Cys Asn Ala Arg Lys Thr Asp Lys Glu Tyr Glu Leu Ile
245 250 255
Asp Ala Leu Ala Lys Lys Leu Gly Thr Gln Met Ile His Phe Val Pro
260 265 270
Arg Asp Asn Gln Val Gln Arg Ala Glu Leu Arg Arg Met Thr Val Ile
275 280 285
Glu Tyr Ser Pro Glu His Pro Gln Ala Gln Glu Tyr Arg Thr Leu Ala
290 295 300
Gln Lys Ile Ala Asp Asn Lys Met Leu Val Val Pro Thr Pro Leu Glu
305 310 315 320
Met Glu Glu Leu Glu Asp Leu Leu Met Glu Phe Gly Ile Met Glu Ala
325 330 335
Glu Asp Glu Ser Ile Val Gly Val Ala Glu Ala Ala Ala Val
340 345 350
<210> 176
<211> 355
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-
CoxIV::TwinStrep::NifH, кодированная SN186. Аминокислоты 1-31
соответствуют последовательности MTP-CoxIV, аминокислоты 32-61
соответствуют последовательности TwinStrep включающей GG на ее C-
конце, и аминокислоты 62-355
<400> 176
Met Leu Ser Leu Arg Gln Ser Ile Arg Phe Phe Lys Pro Ala Thr Arg
1. 5 10 15
Thr Leu Cys Ser Ser Arg Tyr Leu Leu Gln Gln Lys Pro Ser Ala Trp
20 25 30
Ser His Pro Gln Phe Glu Lys Gly Gly Gly Ser Gly Gly Gly Ser Gly
35 40 45
Gly Ser Ala Trp Ser His Pro Gln Phe Glu Lys Gly Gly Met Ala Ser
50 55 60
Leu Arg Gln Ile Ala Phe Tyr Gly Lys Gly Gly Ile Gly Lys Ser Thr
65 70 75 80
Thr Ser Gln Asn Thr Leu Ala Ala Leu Ala Glu Met Gly Gln Lys Ile
85 90 95
Leu Ile Val Gly Cys Asp Pro Lys Ala Asp Ser Thr Arg Leu Ile Leu
100 105 110
His Ala Lys Ala Gln Asp Thr Ile Leu Ser Leu Ala Ala Ser Ala Gly
115 120 125
Ser Val Glu Asp Leu Glu Leu Glu Asp Val Met Lys Val Gly Tyr Gln
130 135 140
Asp Ile Arg Cys Val Glu Ser Gly Gly Pro Glu Pro Gly Val Gly Cys
145 150 155 160
Ala Gly Arg Gly Val Ile Thr Ser Ile Asn Phe Leu Glu Glu Asn Gly
165 170 175
Ala Tyr Glu Asn Ile Asp Tyr Val Ser Tyr Asp Val Leu Gly Asp Val
180 185 190
Val Cys Gly Gly Phe Ala Met Pro Ile Arg Glu Asn Lys Ala Gln Glu
195 200 205
Ile Tyr Ile Val Met Ser Gly Glu Met Met Ala Met Tyr Ala Ala Asn
210 215 220
Asn Ile Ser Lys Gly Ile Leu Lys Tyr Ala Asn Ser Gly Gly Val Arg
225 230 235 240
Leu Gly Gly Leu Ile Cys Asn Glu Arg Gln Thr Asp Lys Glu Leu Glu
245 250 255
Leu Ala Glu Ala Leu Ala Lys Lys Leu Gly Thr Gln Leu Ile Tyr Phe
260 265 270
Val Pro Arg Asp Asn Val Val Gln His Ala Glu Leu Arg Arg Met Thr
275 280 285
Val Leu Glu Tyr Ala Pro Asp Ser Lys Gln Ala Asp His Tyr Arg Lys
290 295 300
Leu Ala Ala Lys Val His Asn Asn Gly Gly Lys Gly Ile Ile Pro Thr
305 310 315 320
Pro Ile Ser Met Asp Glu Leu Glu Asp Met Leu Met Glu His Gly Ile
325 330 335
Ile Lys Ala Val Asp Glu Ser Ile Ile Gly Lys Thr Ala Ala Glu Leu
340 345 350
Ala Ala Ser
355
<210> 177
<211> 336
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-
CoxIV::TwinStrep::NifH, кодированная SN187. Аминокислоты 1-31
соответствуют последовательности MTP-CoxIV, аминокислоты 32-61
соответствуют последовательности TwinStrep включающей GG на ее C-
конце, и аминокислоты 62-336
<400> 177
Met Leu Ser Leu Arg Gln Ser Ile Arg Phe Phe Lys Pro Ala Thr Arg
1. 5 10 15
Thr Leu Cys Ser Ser Arg Tyr Leu Leu Gln Gln Lys Pro Ser Ala Trp
20 25 30
Ser His Pro Gln Phe Glu Lys Gly Gly Gly Ser Gly Gly Gly Ser Gly
35 40 45
Gly Ser Ala Trp Ser His Pro Gln Phe Glu Lys Gly Gly Met Val Arg
50 55 60
Lys Ile Ala Ile Tyr Gly Lys Gly Gly Ile Gly Lys Ser Thr Thr Gln
65 70 75 80
Gln Asn Thr Ala Ala Ala Met Ser Tyr Phe His Gly Lys Asn Val Met
85 90 95
Ile His Gly Cys Asp Pro Lys Ala Asp Ser Thr Arg Leu Ile Leu Gly
100 105 110
Gly Lys Met Gln Thr Thr Met Met Asp Thr Leu Arg Glu Leu Gly Glu
115 120 125
Val Ala Cys Thr Pro Asp Lys Val Ile Glu Thr Gly Phe Gly Gly Ile
130 135 140
Lys Cys Val Glu Ser Gly Gly Pro Glu Pro Gly Val Gly Cys Ala Gly
145 150 155 160
Arg Gly Val Ile Thr Ala Ile Thr Leu Met Glu Arg His Gly Val Tyr
165 170 175
Glu Lys Asp Leu Asp Phe Val Phe Phe Asp Val Leu Gly Asp Val Val
180 185 190
Cys Gly Gly Phe Ala Met Pro Val Arg Asp Gly Lys Ala Glu Glu Ile
195 200 205
Tyr Ile Val Ala Ser Gly Glu Met Met Ala Leu Tyr Ala Ala Asn Asn
210 215 220
Ile Cys Lys Gly Met Val Lys Tyr Ala Arg Gln Ser Gly Val Arg Leu
225 230 235 240
Gly Gly Ile Ile Cys Asn Ser Arg Asn Val Asp Gly Glu Lys Glu Leu
245 250 255
Leu Glu Glu Phe Cys Glu Arg Ile Gly Thr Gln Met Ile His Phe Val
260 265 270
Pro Arg Asp Asn Ile Val Gln Lys Ala Glu Phe Asn Lys Lys Ser Val
275 280 285
Ile Glu Phe Asp Pro Glu Cys Asn Gln Ser Gln Glu Tyr Arg Glu Leu
290 295 300
Ala Arg Lys Ile Ile Glu Asn Lys Asp Phe Val Ile Pro Glu Pro Met
305 310 315 320
Thr Met Asp Glu Met Glu Glu Leu Val Val Lys Tyr Gly Val Met Asp
325 330 335
<210> 178
<211> 334
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-
CoxIV::TwinStrep::NifH, кодированная SN188. Аминокислоты 1-31
соответствуют последовательности MTP-CoxIV, аминокислоты 32-61
соответствуют последовательности TwinStrep включающей GG на ее C-
конце, и аминокислоты 62-334
<400> 178
Met Leu Ser Leu Arg Gln Ser Ile Arg Phe Phe Lys Pro Ala Thr Arg
1. 5 10 15
Thr Leu Cys Ser Ser Arg Tyr Leu Leu Gln Gln Lys Pro Ser Ala Trp
20 25 30
Ser His Pro Gln Phe Glu Lys Gly Gly Gly Ser Gly Gly Gly Ser Gly
35 40 45
Gly Ser Ala Trp Ser His Pro Gln Phe Glu Lys Gly Gly Met Arg Gln
50 55 60
Ile Ala Ile Tyr Gly Lys Gly Gly Ile Gly Lys Ser Thr Thr Thr Gln
65 70 75 80
Asn Leu Thr Ala Ser Leu Ser Thr Met Gly Asn Lys Ile Met Leu Val
85 90 95
Gly Cys Asp Pro Lys Ala Asp Ser Thr Arg Met Leu Leu Gly Gly Leu
100 105 110
Asn Gln Lys Thr Val Leu Asp Thr Leu Arg Ser Glu Gly Asp Glu Gly
115 120 125
Val Asp Leu Asp Val Val Met Gln Arg Gly Phe Gly Asp Ile Lys Cys
130 135 140
Val Glu Ser Gly Gly Pro Glu Pro Gly Val Gly Cys Ala Gly Arg Gly
145 150 155 160
Ile Ile Thr Ser Ile Gly Leu Leu Glu Asn Leu Gly Ala Tyr Thr Asp
165 170 175
Asp Leu Asp Tyr Val Phe Tyr Asp Val Leu Gly Asp Val Val Cys Gly
180 185 190
Gly Phe Ala Met Pro Ile Arg Glu Gly Lys Ala Lys Glu Ile Tyr Ile
195 200 205
Val Ala Ser Gly Glu Leu Met Ala Ile Tyr Ala Ala Asn Asn Ile Cys
210 215 220
Lys Gly Leu Ala Lys Phe Ala Lys Gly Gly Ala Arg Leu Gly Gly Ile
225 230 235 240
Ile Cys Asn Ser Arg Asn Val Asp Gly Glu Arg Glu Leu Leu Asp Ala
245 250 255
Phe Ala Lys Lys Leu Gly Ser His Leu Ile His Phe Ile Pro Arg Asp
260 265 270
Asn Ile Val Gln Arg Ala Glu Ile Asn Arg Lys Thr Val Ile Asp Phe
275 280 285
Asp Pro Glu Ser Asn Gln Ala Lys Glu Tyr Leu Thr Leu Ala His Asn
290 295 300
Val Gln Asn Asn Asp Lys Leu Val Val Pro Thr Pro Leu Pro Met Glu
305 310 315 320
Glu Leu Glu Ala Met Met Val Glu Phe Gly Ile Val Asp Leu
325 330
<210> 179
<211> 336
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-
CoxIV::TwinStrep::NifH, кодированная SN189. Аминокислоты 1-31
соответствуют последовательности MTP-CoxIV, аминокислоты 32-61
соответствуют последовательности TwinStrep включающей GG на ее C-
конце, и аминокислоты 62-336
<400> 179
Met Leu Ser Leu Arg Gln Ser Ile Arg Phe Phe Lys Pro Ala Thr Arg
1. 5 10 15
Thr Leu Cys Ser Ser Arg Tyr Leu Leu Gln Gln Lys Pro Ser Ala Trp
20 25 30
Ser His Pro Gln Phe Glu Lys Gly Gly Gly Ser Gly Gly Gly Ser Gly
35 40 45
Gly Ser Ala Trp Ser His Pro Gln Phe Glu Lys Gly Gly Met Arg Gln
50 55 60
Ile Ala Ile Tyr Gly Lys Gly Gly Ile Gly Lys Ser Thr Thr Thr Gln
65 70 75 80
Asn Thr Val Ala Ala Leu Ala Asp Ala Gly Lys Lys Ile Met Val Val
85 90 95
Gly Cys Asp Pro Lys Ala Asp Ser Thr Arg Leu Leu Leu Asn Gly Leu
100 105 110
Asn Gln Lys Thr Val Leu Asp Thr Leu Arg Asp Glu Gly Glu Asp Val
115 120 125
Ile Leu Glu Asp Val Leu Arg Thr Gly Phe Lys Asp Val Lys Cys Val
130 135 140
Glu Ser Gly Gly Pro Glu Pro Gly Val Gly Cys Ala Gly Arg Gly Ile
145 150 155 160
Ile Thr Ser Ile Asn Leu Leu Glu Ser Leu Gly Ala Tyr Thr Asp Asp
165 170 175
Leu Asp Tyr Val Phe Tyr Asp Val Leu Gly Asp Val Val Cys Gly Gly
180 185 190
Phe Ala Met Pro Ile Arg Glu Gly Lys Ala Arg Glu Ile Tyr Ile Val
195 200 205
Ala Ser Gly Glu Leu Met Ala Leu Tyr Ala Ala Asn Asn Ile Cys Lys
210 215 220
Gly Val Gln Lys Tyr Ala Lys Thr Gly Gly Val Arg Met Gly Gly Ile
225 230 235 240
Ile Cys Asn Ser Arg Lys Val Asp Lys Glu Tyr Asp Leu Leu Lys Ala
245 250 255
Phe Ala Glu Glu Ile Gly Thr Gln Leu Ile His Phe Leu Pro Arg Asp
260 265 270
Asn Val Val Gln Arg Ala Glu Ile Lys Lys Lys Thr Val Ile Asp Tyr
275 280 285
Asp Pro Thr Val Ala Gln Ala Asp Glu Tyr Arg Lys Leu Ala Lys Asn
290 295 300
Ile Asp Glu Asn Thr Met Phe Val Ile Pro Asn Pro Met Thr Gln Asp
305 310 315 320
Arg Leu Glu Glu Leu Met Met Glu His Gly Phe Met Glu Gly Leu Asp
325 330 335
<210> 180
<211> 336
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-
CoxIV::TwinStrep::NifH, кодированная SN190. Аминокислоты 1-31
соответствуют последовательности MTP-CoxIV, аминокислоты 32-61
соответствуют последовательности TwinStrep включающей GG на ее C-
конце, и аминокислоты 62-336
<400> 180
Met Leu Ser Leu Arg Gln Ser Ile Arg Phe Phe Lys Pro Ala Thr Arg
1. 5 10 15
Thr Leu Cys Ser Ser Arg Tyr Leu Leu Gln Gln Lys Pro Ser Ala Trp
20 25 30
Ser His Pro Gln Phe Glu Lys Gly Gly Gly Ser Gly Gly Gly Ser Gly
35 40 45
Gly Ser Ala Trp Ser His Pro Gln Phe Glu Lys Gly Gly Met Thr Arg
50 55 60
Lys Ile Ala Ile Tyr Gly Lys Gly Gly Ile Gly Lys Ser Thr Thr Gln
65 70 75 80
Gln Asn Thr Ala Ala Ala Leu Ala Tyr Phe Tyr Gly Lys Lys Val Leu
85 90 95
Ile His Gly Cys Asp Pro Lys Ala Asp Cys Thr Arg Leu Ile Leu Gly
100 105 110
Gly Lys Pro Gln Glu Thr Val Met Asp Thr Met Arg Glu Leu Gly Glu
115 120 125
Asp Ala Val Thr Ile Asp Arg Val Val Lys Thr Gly Phe Cys Gly Ile
130 135 140
Lys Cys Val Glu Ser Gly Gly Pro Glu Pro Gly Val Gly Cys Ala Gly
145 150 155 160
Arg Gly Val Ile Thr Ala Ile Ser Leu Met Glu Glu Leu Gly Ala Tyr
165 170 175
Thr Pro Asp Leu Asp Phe Ile Phe Phe Asp Val Leu Gly Asp Val Val
180 185 190
Cys Gly Gly Phe Ala Met Pro Val Arg Glu Gly Lys Ala Gln Glu Ile
195 200 205
Tyr Ile Val Ala Ser Gly Glu Met Met Ala Leu Tyr Ala Ala Asn Asn
210 215 220
Ile Cys Arg Gly Met Val Lys Tyr Ala Glu Gln Ser Gly Val Arg Leu
225 230 235 240
Gly Gly Ile Ile Cys Asn Ser Arg Asn Val Asp Gly Glu Arg Glu Leu
245 250 255
Met Glu Glu Phe Cys Ser Lys Ile Gly Thr Gln Met Ile His Phe Ile
260 265 270
Pro Arg Asp Asn Ile Val Gln Lys Ala Glu Phe Asn Arg Gln Thr Val
275 280 285
Thr Gln Phe Asp Pro Asn Cys Asn Gln Ala Gln Glu Tyr Arg Glu Leu
290 295 300
Ala Arg Lys Ile Ile Glu Asn Asp Met Phe Val Ile Pro Lys Pro Met
305 310 315 320
Thr Met Asp Glu Met Glu Asn Leu Val Ile Lys Tyr Gly Leu Leu Glu
325 330 335
<210> 181
<211> 335
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность слитого полипептида MTP-
CoxIV::TwinStrep::NifH, кодированная SN191. Аминокислоты 1-31
соответствуют последовательности MTP-CoxIV, аминокислоты 32-61
соответствуют последовательности TwinStrep включающей GG на ее
C-конце, и аминокислоты 62-335
<400> 181
Met Leu Ser Leu Arg Gln Ser Ile Arg Phe Phe Lys Pro Ala Thr Arg
1. 5 10 15
Thr Leu Cys Ser Ser Arg Tyr Leu Leu Gln Gln Lys Pro Ser Ala Trp
20 25 30
Ser His Pro Gln Phe Glu Lys Gly Gly Gly Ser Gly Gly Gly Ser Gly
35 40 45
Gly Ser Ala Trp Ser His Pro Gln Phe Glu Lys Gly Gly Met Arg Lys
50 55 60
Ile Ala Ile Tyr Gly Arg Gly Gly Val Gly Lys Ser Thr Thr Thr Gln
65 70 75 80
Asn Val Val Ala Gly Leu Ser Glu Met Ser Arg Lys Val Met Val Val
85 90 95
Gly Cys Asp Ser Lys Ala Asp Ser Thr Arg Leu Leu Leu Gly Gly Leu
100 105 110
His Gln Lys Ile Val Leu Asp Thr Leu Arg Lys Glu Glu Asp Asp Val
115 120 125
Asn Leu Glu Asp Phe Arg Leu Glu Gly Trp Gly Lys Thr Leu Cys Val
130 135 140
Glu Ser Gly Gly Pro Glu Pro Gly Val Gly Cys Thr Gly Arg Gly Ile
145 150 155 160
Leu Thr Ser Ile Gly Leu Leu Glu Gln Leu Gly Ala Tyr Asp Asp Ala
165 170 175
Val Arg Leu Asp Tyr Thr Phe Tyr Asp Gly Leu Gly Asp Val Val Cys
180 185 190
Ser Gly Phe Val Met Pro Ile Arg Glu Arg Lys Ala Gln Glu Ile Tyr
195 200 205
Ile Val Thr Ser Gly Glu Ile Met Ala Met Tyr Thr Ala Asn Asn Ile
210 215 220
Cys Arg Ser Leu Gln Lys Tyr Ala Pro Val Gly Gly Ile Arg Leu Gly
225 230 235 240
Gly Leu Ile Cys Asn Ser Arg Lys Val Asp Arg Glu Asn Asp Leu Val
245 250 255
Glu Ala Leu Ala Glu Lys Leu Gly Thr Gln Thr Ile Tyr Ser Ile Pro
260 265 270
Arg Asp Asn Met Val Gln Arg Ala Glu Phe Tyr Arg Lys Thr Val Ile
275 280 285
Glu Tyr Ala Pro Glu Cys Glu Gln Ala Gln His Tyr Arg Asn Leu Ala
290 295 300
Ala Ala Ile Asp Gln Asn Thr Asp Phe Val Ile Pro Lys Ser Met Ser
305 310 315 320
Ser Asp Arg Leu Glu Glu Leu Leu Val Lys Phe Gly Leu Phe Asp
325 330 335
<210> 182
<211> 583
<212> PRT
<213> Искусственная последовательность
<220>
<223> полипептид MTP-FA?51::AnfD::HA
<400> 182
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly Met Pro His His Glu Phe Glu Cys Ser Lys
50 55 60
Val Ile Pro Glu Arg Lys Lys His Ala Val Ile Lys Gly Lys Gly Glu
65 70 75 80
Thr Leu Ala Asp Ala Leu Pro Gln Gly Tyr Leu Asn Thr Ile Pro Gly
85 90 95
Ser Ile Ser Glu Arg Gly Cys Ala Tyr Cys Gly Ala Lys His Val Ile
100 105 110
Gly Thr Pro Met Lys Asp Val Ile His Ile Ser His Gly Pro Val Gly
115 120 125
Cys Thr Tyr Asp Thr Trp Gln Thr Lys Arg Tyr Ile Ser Asp Asn Asp
130 135 140
Asn Phe Gln Leu Lys Tyr Thr Tyr Ala Thr Asp Val Lys Glu Lys His
145 150 155 160
Ile Val Phe Gly Ala Glu Lys Leu Leu Lys Gln Asn Ile Ile Glu Ala
165 170 175
Phe Lys Ala Phe Pro Gln Ile Lys Arg Met Thr Ile Tyr Gln Thr Cys
180 185 190
Ala Thr Ala Leu Ile Gly Asp Asp Ile Asn Ala Ile Ala Glu Glu Val
195 200 205
Met Glu Glu Met Pro Glu Val Asp Ile Phe Val Cys Asn Ser Pro Gly
210 215 220
Phe Ala Gly Pro Ser Gln Ser Gly Gly His His Lys Ile Asn Ile Ala
225 230 235 240
Trp Ile Asn Gln Lys Val Gly Thr Val Glu Pro Glu Ile Thr Gly Asp
245 250 255
His Val Ile Asn Tyr Val Gly Glu Tyr Asn Ile Gln Gly Asp Gln Glu
260 265 270
Val Met Val Asp Tyr Phe Lys Arg Met Gly Ile Gln Val Leu Ser Thr
275 280 285
Phe Thr Gly Asn Gly Ser Tyr Asp Gly Leu Arg Ala Met His Arg Ala
290 295 300
His Leu Asn Val Leu Glu Cys Ala Arg Ser Ala Glu Tyr Ile Cys Asn
305 310 315 320
Glu Leu Arg Val Arg Tyr Gly Ile Pro Arg Leu Asp Ile Asp Gly Phe
325 330 335
Gly Phe Lys Pro Leu Ala Asp Ser Leu Arg Lys Ile Gly Met Phe Phe
340 345 350
Gly Ile Glu Asp Arg Ala Lys Ala Ile Ile Asp Glu Glu Val Ala Arg
355 360 365
Trp Lys Pro Glu Leu Asp Trp Tyr Lys Glu Arg Leu Met Gly Lys Lys
370 375 380
Val Cys Leu Trp Pro Gly Gly Ser Lys Leu Trp His Trp Ala His Val
385 390 395 400
Ile Glu Glu Glu Met Gly Leu Lys Val Val Ser Val Tyr Thr Lys Phe
405 410 415
Gly His Gln Gly Asp Met Glu Lys Gly Ile Ala Arg Cys Gly Glu Gly
420 425 430
Thr Leu Ala Ile Asp Asp Pro Asn Glu Leu Glu Gly Leu Glu Ala Leu
435 440 445
Glu Met Leu Lys Pro Asp Ile Ile Leu Thr Gly Lys Arg Pro Gly Glu
450 455 460
Val Ala Lys Lys Val Arg Val Pro Tyr Leu Asn Ala His Ala Tyr His
465 470 475 480
Asn Gly Pro Tyr Lys Gly Phe Glu Gly Trp Val Arg Phe Ala Arg Asp
485 490 495
Ile Tyr Asn Ala Ile Tyr Ser Pro Ile His Gln Leu Ser Gly Ile Asp
500 505 510
Ile Thr Lys Asp Asn Ala Pro Glu Trp Gly Asn Gly Phe Arg Thr Arg
515 520 525
Gln Met Leu Ser Asp Gly Asn Leu Ser Asp Ala Val Arg Asn Ser Glu
530 535 540
Thr Leu Arg Gln Tyr Thr Gly Gly Tyr Asp Ser Val Ser Lys Leu Arg
545 550 555 560
Glu Arg Glu Tyr Pro Ala Phe Glu Arg Lys Val Gly Gly Gly Tyr Pro
565 570 575
Tyr Asp Val Pro Asp Tyr Ala
580
<210> 183
<211> 530
<212> PRT
<213> Искусственная последовательность
<220>
<223> полипептид HA::AnfD
<400> 183
Met Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Gly Gly Met Pro His His
1. 5 10 15
Glu Phe Glu Cys Ser Lys Val Ile Pro Glu Arg Lys Lys His Ala Val
20 25 30
Ile Lys Gly Lys Gly Glu Thr Leu Ala Asp Ala Leu Pro Gln Gly Tyr
35 40 45
Leu Asn Thr Ile Pro Gly Ser Ile Ser Glu Arg Gly Cys Ala Tyr Cys
50 55 60
Gly Ala Lys His Val Ile Gly Thr Pro Met Lys Asp Val Ile His Ile
65 70 75 80
Ser His Gly Pro Val Gly Cys Thr Tyr Asp Thr Trp Gln Thr Lys Arg
85 90 95
Tyr Ile Ser Asp Asn Asp Asn Phe Gln Leu Lys Tyr Thr Tyr Ala Thr
100 105 110
Asp Val Lys Glu Lys His Ile Val Phe Gly Ala Glu Lys Leu Leu Lys
115 120 125
Gln Asn Ile Ile Glu Ala Phe Lys Ala Phe Pro Gln Ile Lys Arg Met
130 135 140
Thr Ile Tyr Gln Thr Cys Ala Thr Ala Leu Ile Gly Asp Asp Ile Asn
145 150 155 160
Ala Ile Ala Glu Glu Val Met Glu Glu Met Pro Glu Val Asp Ile Phe
165 170 175
Val Cys Asn Ser Pro Gly Phe Ala Gly Pro Ser Gln Ser Gly Gly His
180 185 190
His Lys Ile Asn Ile Ala Trp Ile Asn Gln Lys Val Gly Thr Val Glu
195 200 205
Pro Glu Ile Thr Gly Asp His Val Ile Asn Tyr Val Gly Glu Tyr Asn
210 215 220
Ile Gln Gly Asp Gln Glu Val Met Val Asp Tyr Phe Lys Arg Met Gly
225 230 235 240
Ile Gln Val Leu Ser Thr Phe Thr Gly Asn Gly Ser Tyr Asp Gly Leu
245 250 255
Arg Ala Met His Arg Ala His Leu Asn Val Leu Glu Cys Ala Arg Ser
260 265 270
Ala Glu Tyr Ile Cys Asn Glu Leu Arg Val Arg Tyr Gly Ile Pro Arg
275 280 285
Leu Asp Ile Asp Gly Phe Gly Phe Lys Pro Leu Ala Asp Ser Leu Arg
290 295 300
Lys Ile Gly Met Phe Phe Gly Ile Glu Asp Arg Ala Lys Ala Ile Ile
305 310 315 320
Asp Glu Glu Val Ala Arg Trp Lys Pro Glu Leu Asp Trp Tyr Lys Glu
325 330 335
Arg Leu Met Gly Lys Lys Val Cys Leu Trp Pro Gly Gly Ser Lys Leu
340 345 350
Trp His Trp Ala His Val Ile Glu Glu Glu Met Gly Leu Lys Val Val
355 360 365
Ser Val Tyr Thr Lys Phe Gly His Gln Gly Asp Met Glu Lys Gly Ile
370 375 380
Ala Arg Cys Gly Glu Gly Thr Leu Ala Ile Asp Asp Pro Asn Glu Leu
385 390 395 400
Glu Gly Leu Glu Ala Leu Glu Met Leu Lys Pro Asp Ile Ile Leu Thr
405 410 415
Gly Lys Arg Pro Gly Glu Val Ala Lys Lys Val Arg Val Pro Tyr Leu
420 425 430
Asn Ala His Ala Tyr His Asn Gly Pro Tyr Lys Gly Phe Glu Gly Trp
435 440 445
Val Arg Phe Ala Arg Asp Ile Tyr Asn Ala Ile Tyr Ser Pro Ile His
450 455 460
Gln Leu Ser Gly Ile Asp Ile Thr Lys Asp Asn Ala Pro Glu Trp Gly
465 470 475 480
Asn Gly Phe Arg Thr Arg Gln Met Leu Ser Asp Gly Asn Leu Ser Asp
485 490 495
Ala Val Arg Asn Ser Glu Thr Leu Arg Gln Tyr Thr Gly Gly Tyr Asp
500 505 510
Ser Val Ser Lys Leu Arg Glu Arg Glu Tyr Pro Ala Phe Glu Arg Lys
515 520 525
Val Gly
530
<210> 184
<211> 526
<212> PRT
<213> Искусственная последовательность
<220>
<223> полипептид MTP-FA?51::HA::AnfK
<400> 184
Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro Ser
1. 5 10 15
Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser Asp
20 25 30
Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val Val
35 40 45
Arg Asn Arg Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Gly Gly
50 55 60
Met Thr Cys Glu Val Lys Glu Lys Gly Arg Val Gly Thr Ile Asn Pro
65 70 75 80
Ile Phe Thr Cys Gln Pro Ala Gly Ala Gln Phe Val Ser Ile Gly Ile
85 90 95
Lys Asp Cys Ile Gly Ile Val His Gly Gly Gln Gly Cys Val Met Phe
100 105 110
Val Arg Leu Ile Phe Ser Gln His Tyr Lys Glu Ser Phe Glu Leu Ala
115 120 125
Ser Ser Ser Leu His Glu Asp Gly Ala Val Phe Gly Ala Cys Gly Arg
130 135 140
Val Glu Glu Ala Val Asp Val Leu Leu Ser Arg Tyr Pro Asp Val Lys
145 150 155 160
Val Val Pro Ile Ile Thr Thr Cys Ser Thr Glu Ile Ile Gly Asp Asp
165 170 175
Val Asp Gly Val Ile Lys Lys Leu Asn Glu Gly Leu Leu Lys Glu Lys
180 185 190
Phe Pro Asp Arg Glu Val His Leu Ile Ala Met His Thr Pro Ser Phe
195 200 205
Val Gly Ser Met Ile Ser Gly Tyr Asp Val Ala Val Arg Asp Val Val
210 215 220
Arg His Phe Ala Lys Arg Glu Ala Pro Asn Asp Lys Ile Asn Leu Leu
225 230 235 240
Thr Gly Trp Val Asn Pro Gly Asp Val Lys Glu Leu Lys His Leu Leu
245 250 255
Gly Glu Met Asp Ile Glu Ala Asn Val Leu Phe Glu Ile Glu Ser Phe
260 265 270
Asp Ser Pro Ile Leu Pro Asp Gly Ser Ala Val Ser His Gly Asn Thr
275 280 285
Thr Ile Glu Asp Leu Ile Asp Thr Gly Asn Ala Arg Ala Thr Phe Ala
290 295 300
Leu Asn Arg Tyr Glu Gly Thr Lys Ala Ala Glu Tyr Leu Gln Lys Lys
305 310 315 320
Phe Glu Ile Pro Ala Ile Ile Gly Pro Thr Pro Ile Gly Ile Arg Asn
325 330 335
Thr Asp Ile Phe Leu Gln Asn Leu Lys Lys Ala Thr Gly Lys Pro Ile
340 345 350
Pro Gln Ser Leu Ala His Glu Arg Gly Val Ala Ile Asp Ala Leu Ala
355 360 365
Asp Leu Thr His Met Phe Leu Ala Glu Lys Arg Val Ala Ile Tyr Gly
370 375 380
Ala Pro Asp Leu Val Ile Gly Leu Ala Glu Phe Cys Leu Asp Leu Glu
385 390 395 400
Met Lys Pro Val Leu Leu Leu Leu Gly Asp Asp Asn Ser Lys Tyr Val
405 410 415
Asp Asp Pro Arg Ile Lys Ala Leu Gln Glu Asn Val Asp Tyr Gly Met
420 425 430
Glu Ile Val Thr Asn Ala Asp Phe Trp Glu Leu Glu Asn Arg Ile Lys
435 440 445
Asn Glu Gly Leu Glu Leu Asp Leu Ile Leu Gly His Ser Lys Gly Arg
450 455 460
Phe Ile Ser Ile Asp Tyr Asn Ile Pro Met Leu Arg Val Gly Phe Pro
465 470 475 480
Thr Tyr Asp Arg Ala Gly Leu Phe Arg Tyr Pro Thr Val Gly Tyr Gly
485 490 495
Gly Ala Ile Trp Leu Ala Glu Gln Met Ala Asn Thr Leu Phe Ala Asp
500 505 510
Met Glu His Lys Lys Asn Lys Glu Trp Val Leu Asn Val Trp
515 520 525
<210> 185
<211> 339
<212> PRT
<213> Искусственная последовательность
<220>
<223> полипептид MTP-FA?51::HA::AnfH
<400> 185
Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro Ser
1. 5 10 15
Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser Asp
20 25 30
Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val Val
35 40 45
Arg Asn Arg Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Gly Gly
50 55 60
Met Thr Arg Lys Val Ala Ile Tyr Gly Lys Gly Gly Ile Gly Lys Ser
65 70 75 80
Thr Thr Thr Gln Asn Thr Ala Ala Ala Leu Ala Tyr Phe His Asp Lys
85 90 95
Lys Val Phe Ile His Gly Cys Asp Pro Lys Ala Asp Ser Thr Arg Leu
100 105 110
Ile Leu Gly Gly Lys Pro Gln Glu Thr Leu Met Asp Met Leu Arg Asp
115 120 125
Lys Gly Ala Glu Lys Ile Thr Asn Asp Asp Val Ile Lys Lys Gly Phe
130 135 140
Leu Asp Ile Gln Cys Val Glu Ser Gly Gly Pro Glu Pro Gly Val Gly
145 150 155 160
Cys Ala Gly Arg Gly Val Ile Thr Ala Ile Asp Leu Met Glu Glu Asn
165 170 175
Gly Ala Tyr Thr Asp Asp Leu Asp Phe Val Phe Phe Asp Val Leu Gly
180 185 190
Asp Val Val Cys Gly Gly Phe Ala Met Pro Ile Arg Asp Gly Lys Ala
195 200 205
Gln Glu Val Tyr Ile Val Ala Ser Gly Glu Met Met Ala Ile Tyr Ala
210 215 220
Ala Asn Asn Ile Cys Lys Gly Leu Val Lys Tyr Ala Lys Gln Ser Gly
225 230 235 240
Val Arg Leu Gly Gly Ile Ile Cys Asn Ser Arg Lys Val Asp Gly Glu
245 250 255
Arg Glu Phe Leu Glu Glu Phe Thr Ala Ala Ile Gly Thr Lys Met Ile
260 265 270
His Phe Val Pro Arg Asp Asn Ile Val Gln Lys Ala Glu Phe Asn Lys
275 280 285
Lys Thr Val Thr Glu Phe Ala Pro Glu Glu Asn Gln Ala Lys Glu Tyr
290 295 300
Gly Glu Leu Ala Arg Lys Ile Ile Glu Asn Asp Glu Phe Val Ile Pro
305 310 315 320
Lys Pro Leu Thr Met Asp Gln Leu Glu Asp Met Val Val Lys Tyr Gly
325 330 335
Ile Ala Asp
<210> 186
<211> 196
<212> PRT
<213> Искусственная последовательность
<220>
<223> полипептид MTP-FA?51::HA::AnfG
<400> 186
Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro Ser
1. 5 10 15
Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser Asp
20 25 30
Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val Val
35 40 45
Arg Asn Arg Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Gly Gly
50 55 60
Met Ser Thr Ala Ser Ala Ala Ala Val Val Lys Gln Lys Val Glu Ala
65 70 75 80
Pro Val His Pro Met Asp Ala Arg Ile Asp Glu Leu Thr Asp Tyr Ile
85 90 95
Met Lys Asn Cys Leu Trp Gln Phe His Ser Arg Ser Trp Asp Arg Glu
100 105 110
Arg Gln Asn Ala Glu Ile Leu Lys Lys Thr Lys Glu Leu Leu Cys Gly
115 120 125
Glu Pro Val Asp Leu Ser Thr Ser His Asp Arg Cys Tyr Trp Val Asp
130 135 140
Ala Val Cys Leu Ala Asp Asp Tyr Arg Glu His Tyr Pro Trp Ile Asn
145 150 155 160
Ser Met Ser Lys Glu Glu Ile Gly Ser Leu Met Gln Gly Leu Lys Asp
165 170 175
Arg Met Asp Tyr Leu Thr Ile Thr Gly Ser Leu Asn Glu Glu Leu Ser
180 185 190
Asp Lys His Tyr
195
<210> 187
<211> 474
<212> PRT
<213> Искусственная последовательность
<220>
<223> полипептид HA::AnfK
<400> 187
Met Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Gly Gly Met Thr Cys Glu
1. 5 10 15
Val Lys Glu Lys Gly Arg Val Gly Thr Ile Asn Pro Ile Phe Thr Cys
20 25 30
Gln Pro Ala Gly Ala Gln Phe Val Ser Ile Gly Ile Lys Asp Cys Ile
35 40 45
Gly Ile Val His Gly Gly Gln Gly Cys Val Met Phe Val Arg Leu Ile
50 55 60
Phe Ser Gln His Tyr Lys Glu Ser Phe Glu Leu Ala Ser Ser Ser Leu
65 70 75 80
His Glu Asp Gly Ala Val Phe Gly Ala Cys Gly Arg Val Glu Glu Ala
85 90 95
Val Asp Val Leu Leu Ser Arg Tyr Pro Asp Val Lys Val Val Pro Ile
100 105 110
Ile Thr Thr Cys Ser Thr Glu Ile Ile Gly Asp Asp Val Asp Gly Val
115 120 125
Ile Lys Lys Leu Asn Glu Gly Leu Leu Lys Glu Lys Phe Pro Asp Arg
130 135 140
Glu Val His Leu Ile Ala Met His Thr Pro Ser Phe Val Gly Ser Met
145 150 155 160
Ile Ser Gly Tyr Asp Val Ala Val Arg Asp Val Val Arg His Phe Ala
165 170 175
Lys Arg Glu Ala Pro Asn Asp Lys Ile Asn Leu Leu Thr Gly Trp Val
180 185 190
Asn Pro Gly Asp Val Lys Glu Leu Lys His Leu Leu Gly Glu Met Asp
195 200 205
Ile Glu Ala Asn Val Leu Phe Glu Ile Glu Ser Phe Asp Ser Pro Ile
210 215 220
Leu Pro Asp Gly Ser Ala Val Ser His Gly Asn Thr Thr Ile Glu Asp
225 230 235 240
Leu Ile Asp Thr Gly Asn Ala Arg Ala Thr Phe Ala Leu Asn Arg Tyr
245 250 255
Glu Gly Thr Lys Ala Ala Glu Tyr Leu Gln Lys Lys Phe Glu Ile Pro
260 265 270
Ala Ile Ile Gly Pro Thr Pro Ile Gly Ile Arg Asn Thr Asp Ile Phe
275 280 285
Leu Gln Asn Leu Lys Lys Ala Thr Gly Lys Pro Ile Pro Gln Ser Leu
290 295 300
Ala His Glu Arg Gly Val Ala Ile Asp Ala Leu Ala Asp Leu Thr His
305 310 315 320
Met Phe Leu Ala Glu Lys Arg Val Ala Ile Tyr Gly Ala Pro Asp Leu
325 330 335
Val Ile Gly Leu Ala Glu Phe Cys Leu Asp Leu Glu Met Lys Pro Val
340 345 350
Leu Leu Leu Leu Gly Asp Asp Asn Ser Lys Tyr Val Asp Asp Pro Arg
355 360 365
Ile Lys Ala Leu Gln Glu Asn Val Asp Tyr Gly Met Glu Ile Val Thr
370 375 380
Asn Ala Asp Phe Trp Glu Leu Glu Asn Arg Ile Lys Asn Glu Gly Leu
385 390 395 400
Glu Leu Asp Leu Ile Leu Gly His Ser Lys Gly Arg Phe Ile Ser Ile
405 410 415
Asp Tyr Asn Ile Pro Met Leu Arg Val Gly Phe Pro Thr Tyr Asp Arg
420 425 430
Ala Gly Leu Phe Arg Tyr Pro Thr Val Gly Tyr Gly Gly Ala Ile Trp
435 440 445
Leu Ala Glu Gln Met Ala Asn Thr Leu Phe Ala Asp Met Glu His Lys
450 455 460
Lys Asn Lys Glu Trp Val Leu Asn Val Trp
465 470
<210> 188
<211> 287
<212> PRT
<213> Искусственная последовательность
<220>
<223> полипептид HA::AnfH
<400> 188
Met Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Gly Gly Met Thr Arg Lys
1. 5 10 15
Val Ala Ile Tyr Gly Lys Gly Gly Ile Gly Lys Ser Thr Thr Thr Gln
20 25 30
Asn Thr Ala Ala Ala Leu Ala Tyr Phe His Asp Lys Lys Val Phe Ile
35 40 45
His Gly Cys Asp Pro Lys Ala Asp Ser Thr Arg Leu Ile Leu Gly Gly
50 55 60
Lys Pro Gln Glu Thr Leu Met Asp Met Leu Arg Asp Lys Gly Ala Glu
65 70 75 80
Lys Ile Thr Asn Asp Asp Val Ile Lys Lys Gly Phe Leu Asp Ile Gln
85 90 95
Cys Val Glu Ser Gly Gly Pro Glu Pro Gly Val Gly Cys Ala Gly Arg
100 105 110
Gly Val Ile Thr Ala Ile Asp Leu Met Glu Glu Asn Gly Ala Tyr Thr
115 120 125
Asp Asp Leu Asp Phe Val Phe Phe Asp Val Leu Gly Asp Val Val Cys
130 135 140
Gly Gly Phe Ala Met Pro Ile Arg Asp Gly Lys Ala Gln Glu Val Tyr
145 150 155 160
Ile Val Ala Ser Gly Glu Met Met Ala Ile Tyr Ala Ala Asn Asn Ile
165 170 175
Cys Lys Gly Leu Val Lys Tyr Ala Lys Gln Ser Gly Val Arg Leu Gly
180 185 190
Gly Ile Ile Cys Asn Ser Arg Lys Val Asp Gly Glu Arg Glu Phe Leu
195 200 205
Glu Glu Phe Thr Ala Ala Ile Gly Thr Lys Met Ile His Phe Val Pro
210 215 220
Arg Asp Asn Ile Val Gln Lys Ala Glu Phe Asn Lys Lys Thr Val Thr
225 230 235 240
Glu Phe Ala Pro Glu Glu Asn Gln Ala Lys Glu Tyr Gly Glu Leu Ala
245 250 255
Arg Lys Ile Ile Glu Asn Asp Glu Phe Val Ile Pro Lys Pro Leu Thr
260 265 270
Met Asp Gln Leu Glu Asp Met Val Val Lys Tyr Gly Ile Ala Asp
275 280 285
<210> 189
<211> 144
<212> PRT
<213> Искусственная последовательность
<220>
<223> полипептид HA::AnfG
<400> 189
Met Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Gly Gly Met Ser Thr Ala
1. 5 10 15
Ser Ala Ala Ala Val Val Lys Gln Lys Val Glu Ala Pro Val His Pro
20 25 30
Met Asp Ala Arg Ile Asp Glu Leu Thr Asp Tyr Ile Met Lys Asn Cys
35 40 45
Leu Trp Gln Phe His Ser Arg Ser Trp Asp Arg Glu Arg Gln Asn Ala
50 55 60
Glu Ile Leu Lys Lys Thr Lys Glu Leu Leu Cys Gly Glu Pro Val Asp
65 70 75 80
Leu Ser Thr Ser His Asp Arg Cys Tyr Trp Val Asp Ala Val Cys Leu
85 90 95
Ala Asp Asp Tyr Arg Glu His Tyr Pro Trp Ile Asn Ser Met Ser Lys
100 105 110
Glu Glu Ile Gly Ser Leu Met Gln Gly Leu Lys Asp Arg Met Asp Tyr
115 120 125
Leu Thr Ile Thr Gly Ser Leu Asn Glu Glu Leu Ser Asp Lys His Tyr
130 135 140
<210> 190
<211> 526
<212> PRT
<213> Искусственная последовательность
<220>
<223> полипептид mFA?51::HA::AnfK
<400> 190
Met Ala Met Ala Val Phe Arg Arg Glu Ala Ala Ala Leu Leu Pro Ser
1. 5 10 15
Ile Ala Ala Arg Pro Ile Ala Ala Ala Ala Ala Ala Ala Ser Ser Asp
20 25 30
Gln Glu Glu Gly Leu Leu Ala Ala Ala Ala Ala Ala Ala Ala Val Val
35 40 45
Arg Asn Arg Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Gly Gly
50 55 60
Met Thr Cys Glu Val Lys Glu Lys Gly Arg Val Gly Thr Ile Asn Pro
65 70 75 80
Ile Phe Thr Cys Gln Pro Ala Gly Ala Gln Phe Val Ser Ile Gly Ile
85 90 95
Lys Asp Cys Ile Gly Ile Val His Gly Gly Gln Gly Cys Val Met Phe
100 105 110
Val Arg Leu Ile Phe Ser Gln His Tyr Lys Glu Ser Phe Glu Leu Ala
115 120 125
Ser Ser Ser Leu His Glu Asp Gly Ala Val Phe Gly Ala Cys Gly Arg
130 135 140
Val Glu Glu Ala Val Asp Val Leu Leu Ser Arg Tyr Pro Asp Val Lys
145 150 155 160
Val Val Pro Ile Ile Thr Thr Cys Ser Thr Glu Ile Ile Gly Asp Asp
165 170 175
Val Asp Gly Val Ile Lys Lys Leu Asn Glu Gly Leu Leu Lys Glu Lys
180 185 190
Phe Pro Asp Arg Glu Val His Leu Ile Ala Met His Thr Pro Ser Phe
195 200 205
Val Gly Ser Met Ile Ser Gly Tyr Asp Val Ala Val Arg Asp Val Val
210 215 220
Arg His Phe Ala Lys Arg Glu Ala Pro Asn Asp Lys Ile Asn Leu Leu
225 230 235 240
Thr Gly Trp Val Asn Pro Gly Asp Val Lys Glu Leu Lys His Leu Leu
245 250 255
Gly Glu Met Asp Ile Glu Ala Asn Val Leu Phe Glu Ile Glu Ser Phe
260 265 270
Asp Ser Pro Ile Leu Pro Asp Gly Ser Ala Val Ser His Gly Asn Thr
275 280 285
Thr Ile Glu Asp Leu Ile Asp Thr Gly Asn Ala Arg Ala Thr Phe Ala
290 295 300
Leu Asn Arg Tyr Glu Gly Thr Lys Ala Ala Glu Tyr Leu Gln Lys Lys
305 310 315 320
Phe Glu Ile Pro Ala Ile Ile Gly Pro Thr Pro Ile Gly Ile Arg Asn
325 330 335
Thr Asp Ile Phe Leu Gln Asn Leu Lys Lys Ala Thr Gly Lys Pro Ile
340 345 350
Pro Gln Ser Leu Ala His Glu Arg Gly Val Ala Ile Asp Ala Leu Ala
355 360 365
Asp Leu Thr His Met Phe Leu Ala Glu Lys Arg Val Ala Ile Tyr Gly
370 375 380
Ala Pro Asp Leu Val Ile Gly Leu Ala Glu Phe Cys Leu Asp Leu Glu
385 390 395 400
Met Lys Pro Val Leu Leu Leu Leu Gly Asp Asp Asn Ser Lys Tyr Val
405 410 415
Asp Asp Pro Arg Ile Lys Ala Leu Gln Glu Asn Val Asp Tyr Gly Met
420 425 430
Glu Ile Val Thr Asn Ala Asp Phe Trp Glu Leu Glu Asn Arg Ile Lys
435 440 445
Asn Glu Gly Leu Glu Leu Asp Leu Ile Leu Gly His Ser Lys Gly Arg
450 455 460
Phe Ile Ser Ile Asp Tyr Asn Ile Pro Met Leu Arg Val Gly Phe Pro
465 470 475 480
Thr Tyr Asp Arg Ala Gly Leu Phe Arg Tyr Pro Thr Val Gly Tyr Gly
485 490 495
Gly Ala Ile Trp Leu Ala Glu Gln Met Ala Asn Thr Leu Phe Ala Asp
500 505 510
Met Glu His Lys Lys Asn Lys Glu Trp Val Leu Asn Val Trp
515 520 525
<210> 191
<211> 339
<212> PRT
<213> Искусственная последовательность
<220>
<223> полипептид mFA?51::HA::AnfH
<400> 191
Met Ala Met Ala Val Phe Arg Arg Glu Ala Ala Ala Leu Leu Pro Ser
1. 5 10 15
Ile Ala Ala Arg Pro Ile Ala Ala Ala Ala Ala Ala Ala Ser Ser Asp
20 25 30
Gln Glu Glu Gly Leu Leu Ala Ala Ala Ala Ala Ala Ala Ala Val Val
35 40 45
Arg Asn Arg Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Gly Gly
50 55 60
Met Thr Arg Lys Val Ala Ile Tyr Gly Lys Gly Gly Ile Gly Lys Ser
65 70 75 80
Thr Thr Thr Gln Asn Thr Ala Ala Ala Leu Ala Tyr Phe His Asp Lys
85 90 95
Lys Val Phe Ile His Gly Cys Asp Pro Lys Ala Asp Ser Thr Arg Leu
100 105 110
Ile Leu Gly Gly Lys Pro Gln Glu Thr Leu Met Asp Met Leu Arg Asp
115 120 125
Lys Gly Ala Glu Lys Ile Thr Asn Asp Asp Val Ile Lys Lys Gly Phe
130 135 140
Leu Asp Ile Gln Cys Val Glu Ser Gly Gly Pro Glu Pro Gly Val Gly
145 150 155 160
Cys Ala Gly Arg Gly Val Ile Thr Ala Ile Asp Leu Met Glu Glu Asn
165 170 175
Gly Ala Tyr Thr Asp Asp Leu Asp Phe Val Phe Phe Asp Val Leu Gly
180 185 190
Asp Val Val Cys Gly Gly Phe Ala Met Pro Ile Arg Asp Gly Lys Ala
195 200 205
Gln Glu Val Tyr Ile Val Ala Ser Gly Glu Met Met Ala Ile Tyr Ala
210 215 220
Ala Asn Asn Ile Cys Lys Gly Leu Val Lys Tyr Ala Lys Gln Ser Gly
225 230 235 240
Val Arg Leu Gly Gly Ile Ile Cys Asn Ser Arg Lys Val Asp Gly Glu
245 250 255
Arg Glu Phe Leu Glu Glu Phe Thr Ala Ala Ile Gly Thr Lys Met Ile
260 265 270
His Phe Val Pro Arg Asp Asn Ile Val Gln Lys Ala Glu Phe Asn Lys
275 280 285
Lys Thr Val Thr Glu Phe Ala Pro Glu Glu Asn Gln Ala Lys Glu Tyr
290 295 300
Gly Glu Leu Ala Arg Lys Ile Ile Glu Asn Asp Glu Phe Val Ile Pro
305 310 315 320
Lys Pro Leu Thr Met Asp Gln Leu Glu Asp Met Val Val Lys Tyr Gly
325 330 335
Ile Ala Asp
<210> 192
<211> 196
<212> PRT
<213> Искусственная последовательность
<220>
<223> полипептид mFA?51::HA::AnfG
<400> 192
Met Ala Met Ala Val Phe Arg Arg Glu Ala Ala Ala Leu Leu Pro Ser
1. 5 10 15
Ile Ala Ala Arg Pro Ile Ala Ala Ala Ala Ala Ala Ala Ser Ser Asp
20 25 30
Gln Glu Glu Gly Leu Leu Ala Ala Ala Ala Ala Ala Ala Ala Val Val
35 40 45
Arg Asn Arg Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Gly Gly
50 55 60
Met Ser Thr Ala Ser Ala Ala Ala Val Val Lys Gln Lys Val Glu Ala
65 70 75 80
Pro Val His Pro Met Asp Ala Arg Ile Asp Glu Leu Thr Asp Tyr Ile
85 90 95
Met Lys Asn Cys Leu Trp Gln Phe His Ser Arg Ser Trp Asp Arg Glu
100 105 110
Arg Gln Asn Ala Glu Ile Leu Lys Lys Thr Lys Glu Leu Leu Cys Gly
115 120 125
Glu Pro Val Asp Leu Ser Thr Ser His Asp Arg Cys Tyr Trp Val Asp
130 135 140
Ala Val Cys Leu Ala Asp Asp Tyr Arg Glu His Tyr Pro Trp Ile Asn
145 150 155 160
Ser Met Ser Lys Glu Glu Ile Gly Ser Leu Met Gln Gly Leu Lys Asp
165 170 175
Arg Met Asp Tyr Leu Thr Ile Thr Gly Ser Leu Asn Glu Glu Leu Ser
180 185 190
Asp Lys His Tyr
195
<210> 193
<211> 582
<212> PRT
<213> Искусственная последовательность
<220>
<223> полипептид mFA?51::HA::AnfD
<400> 193
Met Ala Met Ala Val Phe Arg Arg Glu Ala Ala Ala Leu Leu Pro Ser
1. 5 10 15
Ile Ala Ala Arg Pro Ile Ala Ala Ala Ala Ala Ala Ala Ser Ser Asp
20 25 30
Gln Glu Glu Gly Leu Leu Ala Ala Ala Ala Ala Ala Ala Ala Val Val
35 40 45
Arg Asn Arg Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Gly Gly
50 55 60
Met Pro His His Glu Phe Glu Cys Ser Lys Val Ile Pro Glu Arg Lys
65 70 75 80
Lys His Ala Val Ile Lys Gly Lys Gly Glu Thr Leu Ala Asp Ala Leu
85 90 95
Pro Gln Gly Tyr Leu Asn Thr Ile Pro Gly Ser Ile Ser Glu Arg Gly
100 105 110
Cys Ala Tyr Cys Gly Ala Lys His Val Ile Gly Thr Pro Met Lys Asp
115 120 125
Val Ile His Ile Ser His Gly Pro Val Gly Cys Thr Tyr Asp Thr Trp
130 135 140
Gln Thr Lys Arg Tyr Ile Ser Asp Asn Asp Asn Phe Gln Leu Lys Tyr
145 150 155 160
Thr Tyr Ala Thr Asp Val Lys Glu Lys His Ile Val Phe Gly Ala Glu
165 170 175
Lys Leu Leu Lys Gln Asn Ile Ile Glu Ala Phe Lys Ala Phe Pro Gln
180 185 190
Ile Lys Arg Met Thr Ile Tyr Gln Thr Cys Ala Thr Ala Leu Ile Gly
195 200 205
Asp Asp Ile Asn Ala Ile Ala Glu Glu Val Met Glu Glu Met Pro Glu
210 215 220
Val Asp Ile Phe Val Cys Asn Ser Pro Gly Phe Ala Gly Pro Ser Gln
225 230 235 240
Ser Gly Gly His His Lys Ile Asn Ile Ala Trp Ile Asn Gln Lys Val
245 250 255
Gly Thr Val Glu Pro Glu Ile Thr Gly Asp His Val Ile Asn Tyr Val
260 265 270
Gly Glu Tyr Asn Ile Gln Gly Asp Gln Glu Val Met Val Asp Tyr Phe
275 280 285
Lys Arg Met Gly Ile Gln Val Leu Ser Thr Phe Thr Gly Asn Gly Ser
290 295 300
Tyr Asp Gly Leu Arg Ala Met His Arg Ala His Leu Asn Val Leu Glu
305 310 315 320
Cys Ala Arg Ser Ala Glu Tyr Ile Cys Asn Glu Leu Arg Val Arg Tyr
325 330 335
Gly Ile Pro Arg Leu Asp Ile Asp Gly Phe Gly Phe Lys Pro Leu Ala
340 345 350
Asp Ser Leu Arg Lys Ile Gly Met Phe Phe Gly Ile Glu Asp Arg Ala
355 360 365
Lys Ala Ile Ile Asp Glu Glu Val Ala Arg Trp Lys Pro Glu Leu Asp
370 375 380
Trp Tyr Lys Glu Arg Leu Met Gly Lys Lys Val Cys Leu Trp Pro Gly
385 390 395 400
Gly Ser Lys Leu Trp His Trp Ala His Val Ile Glu Glu Glu Met Gly
405 410 415
Leu Lys Val Val Ser Val Tyr Thr Lys Phe Gly His Gln Gly Asp Met
420 425 430
Glu Lys Gly Ile Ala Arg Cys Gly Glu Gly Thr Leu Ala Ile Asp Asp
435 440 445
Pro Asn Glu Leu Glu Gly Leu Glu Ala Leu Glu Met Leu Lys Pro Asp
450 455 460
Ile Ile Leu Thr Gly Lys Arg Pro Gly Glu Val Ala Lys Lys Val Arg
465 470 475 480
Val Pro Tyr Leu Asn Ala His Ala Tyr His Asn Gly Pro Tyr Lys Gly
485 490 495
Phe Glu Gly Trp Val Arg Phe Ala Arg Asp Ile Tyr Asn Ala Ile Tyr
500 505 510
Ser Pro Ile His Gln Leu Ser Gly Ile Asp Ile Thr Lys Asp Asn Ala
515 520 525
Pro Glu Trp Gly Asn Gly Phe Arg Thr Arg Gln Met Leu Ser Asp Gly
530 535 540
Asn Leu Ser Asp Ala Val Arg Asn Ser Glu Thr Leu Arg Gln Tyr Thr
545 550 555 560
Gly Gly Tyr Asp Ser Val Ser Lys Leu Arg Glu Arg Glu Tyr Pro Ala
565 570 575
Phe Glu Arg Lys Val Gly
580
<210> 194
<211> 582
<212> PRT
<213> Искусственная последовательность
<220>
<223> полипептид MTP-FA?51::HA::AnfD
<400> 194
Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro Ser
1. 5 10 15
Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser Asp
20 25 30
Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val Val
35 40 45
Arg Asn Arg Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Gly Gly
50 55 60
Met Pro His His Glu Phe Glu Cys Ser Lys Val Ile Pro Glu Arg Lys
65 70 75 80
Lys His Ala Val Ile Lys Gly Lys Gly Glu Thr Leu Ala Asp Ala Leu
85 90 95
Pro Gln Gly Tyr Leu Asn Thr Ile Pro Gly Ser Ile Ser Glu Arg Gly
100 105 110
Cys Ala Tyr Cys Gly Ala Lys His Val Ile Gly Thr Pro Met Lys Asp
115 120 125
Val Ile His Ile Ser His Gly Pro Val Gly Cys Thr Tyr Asp Thr Trp
130 135 140
Gln Thr Lys Arg Tyr Ile Ser Asp Asn Asp Asn Phe Gln Leu Lys Tyr
145 150 155 160
Thr Tyr Ala Thr Asp Val Lys Glu Lys His Ile Val Phe Gly Ala Glu
165 170 175
Lys Leu Leu Lys Gln Asn Ile Ile Glu Ala Phe Lys Ala Phe Pro Gln
180 185 190
Ile Lys Arg Met Thr Ile Tyr Gln Thr Cys Ala Thr Ala Leu Ile Gly
195 200 205
Asp Asp Ile Asn Ala Ile Ala Glu Glu Val Met Glu Glu Met Pro Glu
210 215 220
Val Asp Ile Phe Val Cys Asn Ser Pro Gly Phe Ala Gly Pro Ser Gln
225 230 235 240
Ser Gly Gly His His Lys Ile Asn Ile Ala Trp Ile Asn Gln Lys Val
245 250 255
Gly Thr Val Glu Pro Glu Ile Thr Gly Asp His Val Ile Asn Tyr Val
260 265 270
Gly Glu Tyr Asn Ile Gln Gly Asp Gln Glu Val Met Val Asp Tyr Phe
275 280 285
Lys Arg Met Gly Ile Gln Val Leu Ser Thr Phe Thr Gly Asn Gly Ser
290 295 300
Tyr Asp Gly Leu Arg Ala Met His Arg Ala His Leu Asn Val Leu Glu
305 310 315 320
Cys Ala Arg Ser Ala Glu Tyr Ile Cys Asn Glu Leu Arg Val Arg Tyr
325 330 335
Gly Ile Pro Arg Leu Asp Ile Asp Gly Phe Gly Phe Lys Pro Leu Ala
340 345 350
Asp Ser Leu Arg Lys Ile Gly Met Phe Phe Gly Ile Glu Asp Arg Ala
355 360 365
Lys Ala Ile Ile Asp Glu Glu Val Ala Arg Trp Lys Pro Glu Leu Asp
370 375 380
Trp Tyr Lys Glu Arg Leu Met Gly Lys Lys Val Cys Leu Trp Pro Gly
385 390 395 400
Gly Ser Lys Leu Trp His Trp Ala His Val Ile Glu Glu Glu Met Gly
405 410 415
Leu Lys Val Val Ser Val Tyr Thr Lys Phe Gly His Gln Gly Asp Met
420 425 430
Glu Lys Gly Ile Ala Arg Cys Gly Glu Gly Thr Leu Ala Ile Asp Asp
435 440 445
Pro Asn Glu Leu Glu Gly Leu Glu Ala Leu Glu Met Leu Lys Pro Asp
450 455 460
Ile Ile Leu Thr Gly Lys Arg Pro Gly Glu Val Ala Lys Lys Val Arg
465 470 475 480
Val Pro Tyr Leu Asn Ala His Ala Tyr His Asn Gly Pro Tyr Lys Gly
485 490 495
Phe Glu Gly Trp Val Arg Phe Ala Arg Asp Ile Tyr Asn Ala Ile Tyr
500 505 510
Ser Pro Ile His Gln Leu Ser Gly Ile Asp Ile Thr Lys Asp Asn Ala
515 520 525
Pro Glu Trp Gly Asn Gly Phe Arg Thr Arg Gln Met Leu Ser Asp Gly
530 535 540
Asn Leu Ser Asp Ala Val Arg Asn Ser Glu Thr Leu Arg Gln Tyr Thr
545 550 555 560
Gly Gly Tyr Asp Ser Val Ser Lys Leu Arg Glu Arg Glu Tyr Pro Ala
565 570 575
Phe Glu Arg Lys Val Gly
580
<210> 195
<211> 604
<212> PRT
<213> Искусственная последовательность
<220>
<223> полипептид MTP-FA?51::AnfD::Twin Strep
<400> 195
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly Met Pro His His Glu Phe Glu Cys Ser Lys
50 55 60
Val Ile Pro Glu Arg Lys Lys His Ala Val Ile Lys Gly Lys Gly Glu
65 70 75 80
Thr Leu Ala Asp Ala Leu Pro Gln Gly Tyr Leu Asn Thr Ile Pro Gly
85 90 95
Ser Ile Ser Glu Arg Gly Cys Ala Tyr Cys Gly Ala Lys His Val Ile
100 105 110
Gly Thr Pro Met Lys Asp Val Ile His Ile Ser His Gly Pro Val Gly
115 120 125
Cys Thr Tyr Asp Thr Trp Gln Thr Lys Arg Tyr Ile Ser Asp Asn Asp
130 135 140
Asn Phe Gln Leu Lys Tyr Thr Tyr Ala Thr Asp Val Lys Glu Lys His
145 150 155 160
Ile Val Phe Gly Ala Glu Lys Leu Leu Lys Gln Asn Ile Ile Glu Ala
165 170 175
Phe Lys Ala Phe Pro Gln Ile Lys Arg Met Thr Ile Tyr Gln Thr Cys
180 185 190
Ala Thr Ala Leu Ile Gly Asp Asp Ile Asn Ala Ile Ala Glu Glu Val
195 200 205
Met Glu Glu Met Pro Glu Val Asp Ile Phe Val Cys Asn Ser Pro Gly
210 215 220
Phe Ala Gly Pro Ser Gln Ser Gly Gly His His Lys Ile Asn Ile Ala
225 230 235 240
Trp Ile Asn Gln Lys Val Gly Thr Val Glu Pro Glu Ile Thr Gly Asp
245 250 255
His Val Ile Asn Tyr Val Gly Glu Tyr Asn Ile Gln Gly Asp Gln Glu
260 265 270
Val Met Val Asp Tyr Phe Lys Arg Met Gly Ile Gln Val Leu Ser Thr
275 280 285
Phe Thr Gly Asn Gly Ser Tyr Asp Gly Leu Arg Ala Met His Arg Ala
290 295 300
His Leu Asn Val Leu Glu Cys Ala Arg Ser Ala Glu Tyr Ile Cys Asn
305 310 315 320
Glu Leu Arg Val Arg Tyr Gly Ile Pro Arg Leu Asp Ile Asp Gly Phe
325 330 335
Gly Phe Lys Pro Leu Ala Asp Ser Leu Arg Lys Ile Gly Met Phe Phe
340 345 350
Gly Ile Glu Asp Arg Ala Lys Ala Ile Ile Asp Glu Glu Val Ala Arg
355 360 365
Trp Lys Pro Glu Leu Asp Trp Tyr Lys Glu Arg Leu Met Gly Lys Lys
370 375 380
Val Cys Leu Trp Pro Gly Gly Ser Lys Leu Trp His Trp Ala His Val
385 390 395 400
Ile Glu Glu Glu Met Gly Leu Lys Val Val Ser Val Tyr Thr Lys Phe
405 410 415
Gly His Gln Gly Asp Met Glu Lys Gly Ile Ala Arg Cys Gly Glu Gly
420 425 430
Thr Leu Ala Ile Asp Asp Pro Asn Glu Leu Glu Gly Leu Glu Ala Leu
435 440 445
Glu Met Leu Lys Pro Asp Ile Ile Leu Thr Gly Lys Arg Pro Gly Glu
450 455 460
Val Ala Lys Lys Val Arg Val Pro Tyr Leu Asn Ala His Ala Tyr His
465 470 475 480
Asn Gly Pro Tyr Lys Gly Phe Glu Gly Trp Val Arg Phe Ala Arg Asp
485 490 495
Ile Tyr Asn Ala Ile Tyr Ser Pro Ile His Gln Leu Ser Gly Ile Asp
500 505 510
Ile Thr Lys Asp Asn Ala Pro Glu Trp Gly Asn Gly Phe Arg Thr Arg
515 520 525
Gln Met Leu Ser Asp Gly Asn Leu Ser Asp Ala Val Arg Asn Ser Glu
530 535 540
Thr Leu Arg Gln Tyr Thr Gly Gly Tyr Asp Ser Val Ser Lys Leu Arg
545 550 555 560
Glu Arg Glu Tyr Pro Ala Phe Glu Arg Lys Val Gly Gly Gly Ser Ala
565 570 575
Trp Ser His Pro Gln Phe Glu Lys Gly Gly Gly Ser Gly Gly Gly Ser
580 585 590
Gly Gly Ser Ala Trp Ser His Pro Gln Phe Glu Lys
595 600
<210> 196
<211> 523
<212> PRT
<213> Искусственная последовательность
<220>
<223> полипептид MTP-CoxIV::TwinStrep::AnfK
<400> 196
Met Leu Ser Leu Arg Gln Ser Ile Arg Phe Phe Lys Pro Ala Thr Arg
1. 5 10 15
Thr Leu Cys Ser Ser Arg Tyr Leu Leu Gln Gln Lys Pro Ser Ala Trp
20 25 30
Ser His Pro Gln Phe Glu Lys Gly Gly Gly Ser Gly Gly Gly Ser Gly
35 40 45
Gly Ser Ala Trp Ser His Pro Gln Phe Glu Lys Gly Gly Met Thr Cys
50 55 60
Glu Val Lys Glu Lys Gly Arg Val Gly Thr Ile Asn Pro Ile Phe Thr
65 70 75 80
Cys Gln Pro Ala Gly Ala Gln Phe Val Ser Ile Gly Ile Lys Asp Cys
85 90 95
Ile Gly Ile Val His Gly Gly Gln Gly Cys Val Met Phe Val Arg Leu
100 105 110
Ile Phe Ser Gln His Tyr Lys Glu Ser Phe Glu Leu Ala Ser Ser Ser
115 120 125
Leu His Glu Asp Gly Ala Val Phe Gly Ala Cys Gly Arg Val Glu Glu
130 135 140
Ala Val Asp Val Leu Leu Ser Arg Tyr Pro Asp Val Lys Val Val Pro
145 150 155 160
Ile Ile Thr Thr Cys Ser Thr Glu Ile Ile Gly Asp Asp Val Asp Gly
165 170 175
Val Ile Lys Lys Leu Asn Glu Gly Leu Leu Lys Glu Lys Phe Pro Asp
180 185 190
Arg Glu Val His Leu Ile Ala Met His Thr Pro Ser Phe Val Gly Ser
195 200 205
Met Ile Ser Gly Tyr Asp Val Ala Val Arg Asp Val Val Arg His Phe
210 215 220
Ala Lys Arg Glu Ala Pro Asn Asp Lys Ile Asn Leu Leu Thr Gly Trp
225 230 235 240
Val Asn Pro Gly Asp Val Lys Glu Leu Lys His Leu Leu Gly Glu Met
245 250 255
Asp Ile Glu Ala Asn Val Leu Phe Glu Ile Glu Ser Phe Asp Ser Pro
260 265 270
Ile Leu Pro Asp Gly Ser Ala Val Ser His Gly Asn Thr Thr Ile Glu
275 280 285
Asp Leu Ile Asp Thr Gly Asn Ala Arg Ala Thr Phe Ala Leu Asn Arg
290 295 300
Tyr Glu Gly Thr Lys Ala Ala Glu Tyr Leu Gln Lys Lys Phe Glu Ile
305 310 315 320
Pro Ala Ile Ile Gly Pro Thr Pro Ile Gly Ile Arg Asn Thr Asp Ile
325 330 335
Phe Leu Gln Asn Leu Lys Lys Ala Thr Gly Lys Pro Ile Pro Gln Ser
340 345 350
Leu Ala His Glu Arg Gly Val Ala Ile Asp Ala Leu Ala Asp Leu Thr
355 360 365
His Met Phe Leu Ala Glu Lys Arg Val Ala Ile Tyr Gly Ala Pro Asp
370 375 380
Leu Val Ile Gly Leu Ala Glu Phe Cys Leu Asp Leu Glu Met Lys Pro
385 390 395 400
Val Leu Leu Leu Leu Gly Asp Asp Asn Ser Lys Tyr Val Asp Asp Pro
405 410 415
Arg Ile Lys Ala Leu Gln Glu Asn Val Asp Tyr Gly Met Glu Ile Val
420 425 430
Thr Asn Ala Asp Phe Trp Glu Leu Glu Asn Arg Ile Lys Asn Glu Gly
435 440 445
Leu Glu Leu Asp Leu Ile Leu Gly His Ser Lys Gly Arg Phe Ile Ser
450 455 460
Ile Asp Tyr Asn Ile Pro Met Leu Arg Val Gly Phe Pro Thr Tyr Asp
465 470 475 480
Arg Ala Gly Leu Phe Arg Tyr Pro Thr Val Gly Tyr Gly Gly Ala Ile
485 490 495
Trp Leu Ala Glu Gln Met Ala Asn Thr Leu Phe Ala Asp Met Glu His
500 505 510
Lys Lys Asn Lys Glu Trp Val Leu Asn Val Trp
515 520
<210> 197
<211> 31
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептидная последовательность
<400> 197
Asn Ser Glu Thr Leu Arg Gln Tyr Thr Gly Gly Tyr Asp Ser Val Ser
1. 5 10 15
Lys Leu Arg Glu Arg Glu Tyr Pro Ala Phe Glu Arg Lys Val Gly
20 25 30
<210> 198
<211> 16
<212> PRT
<213> Искусственная последовательность
<220>
<223> пептидная последовательность
<400> 198
Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Ser
1. 5 10 15
<210> 199
<211> 984
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность полипептида AnfD::линкер16::AnfK
использована для моделирования структуры (Пример 20). Аминокислоты
1-509 соответствуют последовательности AnfD (A. vinelandii) исключая
N-концевой метионин, аминокислоты 510-525 соответствуют
<400> 199
Pro His His Glu Phe Glu Cys Ser Lys Val Ile Pro Glu Arg Lys Lys
1. 5 10 15
His Ala Val Ile Lys Gly Lys Gly Glu Thr Leu Ala Asp Ala Leu Pro
20 25 30
Gln Gly Tyr Leu Asn Thr Ile Pro Gly Ser Ile Ser Glu Arg Gly Cys
35 40 45
Ala Tyr Cys Gly Ala Lys His Val Ile Gly Thr Pro Met Lys Asp Val
50 55 60
Ile His Ile Ser His Gly Pro Val Gly Cys Thr Tyr Asp Thr Trp Gln
65 70 75 80
Thr Lys Arg Tyr Ile Ser Asp Asn Asp Asn Phe Gln Leu Lys Tyr Thr
85 90 95
Tyr Ala Thr Asp Val Lys Glu Lys His Ile Val Phe Gly Ala Glu Lys
100 105 110
Leu Leu Lys Gln Asn Ile Ile Glu Ala Phe Lys Ala Phe Pro Gln Ile
115 120 125
Lys Arg Met Thr Ile Tyr Gln Thr Cys Ala Thr Ala Leu Ile Gly Asp
130 135 140
Asp Ile Asn Ala Ile Ala Glu Glu Val Met Glu Glu Met Pro Glu Val
145 150 155 160
Asp Ile Phe Val Cys Asn Ser Pro Gly Phe Ala Gly Pro Ser Gln Ser
165 170 175
Gly Gly His His Lys Ile Asn Ile Ala Trp Ile Asn Gln Lys Val Gly
180 185 190
Thr Val Glu Pro Glu Ile Thr Gly Asp His Val Ile Asn Tyr Val Gly
195 200 205
Glu Tyr Asn Ile Gln Gly Asp Gln Glu Val Met Val Asp Tyr Phe Lys
210 215 220
Arg Met Gly Ile Gln Val Leu Ser Thr Phe Thr Gly Asn Gly Ser Tyr
225 230 235 240
Asp Gly Leu Arg Ala Met His Arg Ala His Leu Asn Val Leu Glu Cys
245 250 255
Ala Arg Ser Ala Glu Tyr Ile Cys Asn Glu Leu Arg Val Arg Tyr Gly
260 265 270
Ile Pro Arg Leu Asp Ile Asp Gly Phe Gly Phe Lys Pro Leu Ala Asp
275 280 285
Ser Leu Arg Lys Ile Gly Met Phe Phe Gly Ile Glu Asp Arg Ala Lys
290 295 300
Ala Ile Ile Asp Glu Glu Val Ala Arg Trp Lys Pro Glu Leu Asp Trp
305 310 315 320
Tyr Lys Glu Arg Leu Met Gly Lys Lys Val Cys Leu Trp Pro Gly Gly
325 330 335
Ser Lys Leu Trp His Trp Ala His Val Ile Glu Glu Glu Met Gly Leu
340 345 350
Lys Val Val Ser Val Tyr Thr Lys Phe Gly His Gln Gly Asp Met Glu
355 360 365
Lys Gly Ile Ala Arg Cys Gly Glu Gly Thr Leu Ala Ile Asp Asp Pro
370 375 380
Asn Glu Leu Glu Gly Leu Glu Ala Leu Glu Met Leu Lys Pro Asp Ile
385 390 395 400
Ile Leu Thr Gly Lys Arg Pro Gly Glu Val Ala Lys Lys Val Arg Val
405 410 415
Pro Tyr Leu Asn Ala His Ala Tyr His Asn Gly Pro Tyr Lys Gly Phe
420 425 430
Glu Gly Trp Val Arg Phe Ala Arg Asp Ile Tyr Asn Ala Ile Tyr Ser
435 440 445
Pro Ile His Gln Leu Ser Gly Ile Asp Ile Thr Lys Asp Asn Ala Pro
450 455 460
Glu Trp Gly Asn Gly Phe Arg Thr Arg Gln Met Leu Ser Asp Gly Asn
465 470 475 480
Ser Glu Thr Leu Arg Gln Tyr Thr Gly Gly Tyr Asp Ser Val Ser Lys
485 490 495
Leu Arg Glu Arg Glu Tyr Pro Ala Phe Glu Arg Lys Val Gly Gly Gly
500 505 510
Gly Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Ser Val Lys
515 520 525
Glu Lys Gly Arg Val Gly Thr Ile Asn Pro Ile Phe Thr Cys Gln Pro
530 535 540
Ala Gly Ala Gln Phe Val Ser Ile Gly Ile Lys Asp Cys Ile Gly Ile
545 550 555 560
Val His Gly Gly Gln Gly Cys Val Met Phe Val Arg Leu Ile Phe Ser
565 570 575
Gln His Tyr Lys Glu Ser Phe Glu Leu Ala Ser Ser Ser Leu His Glu
580 585 590
Asp Gly Ala Val Phe Gly Ala Cys Gly Arg Val Glu Glu Ala Val Asp
595 600 605
Val Leu Leu Ser Arg Tyr Pro Asp Val Lys Val Val Pro Ile Ile Thr
610 615 620
Thr Cys Ser Thr Glu Ile Ile Gly Asp Asp Val Asp Gly Val Ile Lys
625 630 635 640
Lys Leu Asn Glu Gly Leu Leu Lys Glu Lys Phe Pro Asp Arg Glu Val
645 650 655
His Leu Ile Ala Met His Thr Pro Ser Phe Val Gly Ser Met Ile Ser
660 665 670
Gly Tyr Asp Val Ala Val Arg Asp Val Val Arg His Phe Ala Lys Arg
675 680 685
Glu Ala Pro Asn Asp Lys Ile Asn Leu Leu Thr Gly Trp Val Asn Pro
690 695 700
Gly Asp Val Lys Glu Leu Lys His Leu Leu Gly Glu Met Asp Ile Glu
705 710 715 720
Ala Asn Val Leu Phe Glu Ile Glu Ser Phe Asp Ser Pro Ile Leu Pro
725 730 735
Asp Gly Ser Ala Val Ser His Gly Asn Thr Thr Ile Glu Asp Leu Ile
740 745 750
Asp Thr Gly Asn Ala Arg Ala Thr Phe Ala Leu Asn Arg Tyr Glu Gly
755 760 765
Thr Lys Ala Ala Glu Tyr Leu Gln Lys Lys Phe Glu Ile Pro Ala Ile
770 775 780
Ile Gly Pro Thr Pro Ile Gly Ile Arg Asn Thr Asp Ile Phe Leu Gln
785 790 795 800
Asn Leu Lys Lys Ala Thr Gly Lys Pro Ile Pro Gln Ser Leu Ala His
805 810 815
Glu Arg Gly Val Ala Ile Asp Ala Leu Ala Asp Leu Thr His Met Phe
820 825 830
Leu Ala Glu Lys Arg Val Ala Ile Tyr Gly Ala Pro Asp Leu Val Ile
835 840 845
Gly Leu Ala Glu Phe Cys Leu Asp Leu Glu Met Lys Pro Val Leu Leu
850 855 860
Leu Leu Gly Asp Asp Asn Ser Lys Tyr Val Asp Asp Pro Arg Ile Lys
865 870 875 880
Ala Leu Gln Glu Asn Val Asp Tyr Gly Met Glu Ile Val Thr Asn Ala
885 890 895
Asp Phe Trp Glu Leu Glu Asn Arg Ile Lys Asn Glu Gly Leu Glu Leu
900 905 910
Asp Leu Ile Leu Gly His Ser Lys Gly Arg Phe Ile Ser Ile Asp Tyr
915 920 925
Asn Ile Pro Met Leu Arg Val Gly Phe Pro Thr Tyr Asp Arg Ala Gly
930 935 940
Leu Phe Arg Tyr Pro Thr Val Gly Tyr Gly Gly Ala Ile Trp Leu Ala
945 950 955 960
Glu Gln Met Ala Asn Thr Leu Phe Ala Asp Met Glu His Lys Lys Asn
965 970 975
Lys Glu Trp Val Leu Asn Val Trp
980
<210> 200
<211> 26
<212> PRT
<213> Искусственная последовательность
<220>
<223> Линкерная последовательность
<400> 200
Gly Gly Gly Gly Ser Gly Gly Gly Ser Tyr Pro Tyr Asp Val Pro Asp
1. 5 10 15
Tyr Ala Gly Gly Gly Ser Gly Gly Gly Ser
20 25
<210> 201
<211> 1004
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность полипептида AnfD::линкер26(HA)::AnfK
использована для моделирования структуры. Аминокислоты 1-517
соответствуют последовательности AnfD, аминокислоты 518-543
соответствуют линкеру из 26 аминокислот, а аминокислоты 544-1004
сответствуют AnfK.
<400> 201
Met Pro His His Glu Phe Glu Cys Ser Lys Val Ile Pro Glu Arg Lys
1. 5 10 15
Lys His Ala Val Ile Lys Gly Lys Gly Glu Thr Leu Ala Asp Ala Leu
20 25 30
Pro Gln Gly Tyr Leu Asn Thr Ile Pro Gly Ser Ile Ser Glu Arg Gly
35 40 45
Cys Ala Tyr Cys Gly Ala Lys His Val Ile Gly Thr Pro Met Lys Asp
50 55 60
Val Ile His Ile Ser His Gly Pro Val Gly Cys Thr Tyr Asp Thr Trp
65 70 75 80
Gln Thr Lys Arg Tyr Ile Ser Asp Asn Asp Asn Phe Gln Leu Lys Tyr
85 90 95
Thr Tyr Ala Thr Asp Val Lys Glu Lys His Ile Val Phe Gly Ala Glu
100 105 110
Lys Leu Leu Lys Gln Asn Ile Ile Glu Ala Phe Lys Ala Phe Pro Gln
115 120 125
Ile Lys Arg Met Thr Ile Tyr Gln Thr Cys Ala Thr Ala Leu Ile Gly
130 135 140
Asp Asp Ile Asn Ala Ile Ala Glu Glu Val Met Glu Glu Met Pro Glu
145 150 155 160
Val Asp Ile Phe Val Cys Asn Ser Pro Gly Phe Ala Gly Pro Ser Gln
165 170 175
Ser Gly Gly His His Lys Ile Asn Ile Ala Trp Ile Asn Gln Lys Val
180 185 190
Gly Thr Val Glu Pro Glu Ile Thr Gly Asp His Val Ile Asn Tyr Val
195 200 205
Gly Glu Tyr Asn Ile Gln Gly Asp Gln Glu Val Met Val Asp Tyr Phe
210 215 220
Lys Arg Met Gly Ile Gln Val Leu Ser Thr Phe Thr Gly Asn Gly Ser
225 230 235 240
Tyr Asp Gly Leu Arg Ala Met His Arg Ala His Leu Asn Val Leu Glu
245 250 255
Cys Ala Arg Ser Ala Glu Tyr Ile Cys Asn Glu Leu Arg Val Arg Tyr
260 265 270
Gly Ile Pro Arg Leu Asp Ile Asp Gly Phe Gly Phe Lys Pro Leu Ala
275 280 285
Asp Ser Leu Arg Lys Ile Gly Met Phe Phe Gly Ile Glu Asp Arg Ala
290 295 300
Lys Ala Ile Ile Asp Glu Glu Val Ala Arg Trp Lys Pro Glu Leu Asp
305 310 315 320
Trp Tyr Lys Glu Arg Leu Met Gly Lys Lys Val Cys Leu Trp Pro Gly
325 330 335
Gly Ser Lys Leu Trp His Trp Ala His Val Ile Glu Glu Glu Met Gly
340 345 350
Leu Lys Val Val Ser Val Tyr Thr Lys Phe Gly His Gln Gly Asp Met
355 360 365
Glu Lys Gly Ile Ala Arg Cys Gly Glu Gly Thr Leu Ala Ile Asp Asp
370 375 380
Pro Asn Glu Leu Glu Gly Leu Glu Ala Leu Glu Met Leu Lys Pro Asp
385 390 395 400
Ile Ile Leu Thr Gly Lys Arg Pro Gly Glu Val Ala Lys Lys Val Arg
405 410 415
Val Pro Tyr Leu Asn Ala His Ala Tyr His Asn Gly Pro Tyr Lys Gly
420 425 430
Phe Glu Gly Trp Val Arg Phe Ala Arg Asp Ile Tyr Asn Ala Ile Tyr
435 440 445
Ser Pro Ile His Gln Leu Ser Gly Ile Asp Ile Thr Lys Asp Asn Ala
450 455 460
Pro Glu Trp Gly Asn Gly Phe Arg Thr Arg Gln Met Leu Ser Asp Gly
465 470 475 480
Asn Leu Ser Asp Ala Val Arg Asn Ser Glu Thr Leu Arg Gln Tyr Thr
485 490 495
Gly Gly Tyr Asp Ser Val Ser Lys Leu Arg Glu Arg Glu Tyr Pro Ala
500 505 510
Phe Glu Arg Lys Val Gly Gly Gly Gly Ser Gly Gly Gly Ser Tyr Pro
515 520 525
Tyr Asp Val Pro Asp Tyr Ala Gly Gly Gly Ser Gly Gly Gly Ser Thr
530 535 540
Cys Glu Val Lys Glu Lys Gly Arg Val Gly Thr Ile Asn Pro Ile Phe
545 550 555 560
Thr Cys Gln Pro Ala Gly Ala Gln Phe Val Ser Ile Gly Ile Lys Asp
565 570 575
Cys Ile Gly Ile Val His Gly Gly Gln Gly Cys Val Met Phe Val Arg
580 585 590
Leu Ile Phe Ser Gln His Tyr Lys Glu Ser Phe Glu Leu Ala Ser Ser
595 600 605
Ser Leu His Glu Asp Gly Ala Val Phe Gly Ala Cys Gly Arg Val Glu
610 615 620
Glu Ala Val Asp Val Leu Leu Ser Arg Tyr Pro Asp Val Lys Val Val
625 630 635 640
Pro Ile Ile Thr Thr Cys Ser Thr Glu Ile Ile Gly Asp Asp Val Asp
645 650 655
Gly Val Ile Lys Lys Leu Asn Glu Gly Leu Leu Lys Glu Lys Phe Pro
660 665 670
Asp Arg Glu Val His Leu Ile Ala Met His Thr Pro Ser Phe Val Gly
675 680 685
Ser Met Ile Ser Gly Tyr Asp Val Ala Val Arg Asp Val Val Arg His
690 695 700
Phe Ala Lys Arg Glu Ala Pro Asn Asp Lys Ile Asn Leu Leu Thr Gly
705 710 715 720
Trp Val Asn Pro Gly Asp Val Lys Glu Leu Lys His Leu Leu Gly Glu
725 730 735
Met Asp Ile Glu Ala Asn Val Leu Phe Glu Ile Glu Ser Phe Asp Ser
740 745 750
Pro Ile Leu Pro Asp Gly Ser Ala Val Ser His Gly Asn Thr Thr Ile
755 760 765
Glu Asp Leu Ile Asp Thr Gly Asn Ala Arg Ala Thr Phe Ala Leu Asn
770 775 780
Arg Tyr Glu Gly Thr Lys Ala Ala Glu Tyr Leu Gln Lys Lys Phe Glu
785 790 795 800
Ile Pro Ala Ile Ile Gly Pro Thr Pro Ile Gly Ile Arg Asn Thr Asp
805 810 815
Ile Phe Leu Gln Asn Leu Lys Lys Ala Thr Gly Lys Pro Ile Pro Gln
820 825 830
Ser Leu Ala His Glu Arg Gly Val Ala Ile Asp Ala Leu Ala Asp Leu
835 840 845
Thr His Met Phe Leu Ala Glu Lys Arg Val Ala Ile Tyr Gly Ala Pro
850 855 860
Asp Leu Val Ile Gly Leu Ala Glu Phe Cys Leu Asp Leu Glu Met Lys
865 870 875 880
Pro Val Leu Leu Leu Leu Gly Asp Asp Asn Ser Lys Tyr Val Asp Asp
885 890 895
Pro Arg Ile Lys Ala Leu Gln Glu Asn Val Asp Tyr Gly Met Glu Ile
900 905 910
Val Thr Asn Ala Asp Phe Trp Glu Leu Glu Asn Arg Ile Lys Asn Glu
915 920 925
Gly Leu Glu Leu Asp Leu Ile Leu Gly His Ser Lys Gly Arg Phe Ile
930 935 940
Ser Ile Asp Tyr Asn Ile Pro Met Leu Arg Val Gly Phe Pro Thr Tyr
945 950 955 960
Asp Arg Ala Gly Leu Phe Arg Tyr Pro Thr Val Gly Tyr Gly Gly Ala
965 970 975
Ile Trp Leu Ala Glu Gln Met Ala Asn Thr Leu Phe Ala Asp Met Glu
980 985 990
His Lys Lys Asn Lys Glu Trp Val Leu Asn Val Trp
995 1000
<210> 202
<211> 1068
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность пептида MTP-
FA?51::AnfD::линкер26(HA)::AnfK, кодированная SN272. Аминокислоты 1-64
соответствуют последовательности MTP-FA?51-HA, включая GG на ее
C-конце, аминокислоты 65-581 соответствуют последовательности AnfD
(A. vinelandii),
<400> 202
Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro Ser
1. 5 10 15
Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser Asp
20 25 30
Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val Val
35 40 45
Arg Asn Arg Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Gly Gly
50 55 60
Met Pro His His Glu Phe Glu Cys Ser Lys Val Ile Pro Glu Arg Lys
65 70 75 80
Lys His Ala Val Ile Lys Gly Lys Gly Glu Thr Leu Ala Asp Ala Leu
85 90 95
Pro Gln Gly Tyr Leu Asn Thr Ile Pro Gly Ser Ile Ser Glu Arg Gly
100 105 110
Cys Ala Tyr Cys Gly Ala Lys His Val Ile Gly Thr Pro Met Lys Asp
115 120 125
Val Ile His Ile Ser His Gly Pro Val Gly Cys Thr Tyr Asp Thr Trp
130 135 140
Gln Thr Lys Arg Tyr Ile Ser Asp Asn Asp Asn Phe Gln Leu Lys Tyr
145 150 155 160
Thr Tyr Ala Thr Asp Val Lys Glu Lys His Ile Val Phe Gly Ala Glu
165 170 175
Lys Leu Leu Lys Gln Asn Ile Ile Glu Ala Phe Lys Ala Phe Pro Gln
180 185 190
Ile Lys Arg Met Thr Ile Tyr Gln Thr Cys Ala Thr Ala Leu Ile Gly
195 200 205
Asp Asp Ile Asn Ala Ile Ala Glu Glu Val Met Glu Glu Met Pro Glu
210 215 220
Val Asp Ile Phe Val Cys Asn Ser Pro Gly Phe Ala Gly Pro Ser Gln
225 230 235 240
Ser Gly Gly His His Lys Ile Asn Ile Ala Trp Ile Asn Gln Lys Val
245 250 255
Gly Thr Val Glu Pro Glu Ile Thr Gly Asp His Val Ile Asn Tyr Val
260 265 270
Gly Glu Tyr Asn Ile Gln Gly Asp Gln Glu Val Met Val Asp Tyr Phe
275 280 285
Lys Arg Met Gly Ile Gln Val Leu Ser Thr Phe Thr Gly Asn Gly Ser
290 295 300
Tyr Asp Gly Leu Arg Ala Met His Arg Ala His Leu Asn Val Leu Glu
305 310 315 320
Cys Ala Arg Ser Ala Glu Tyr Ile Cys Asn Glu Leu Arg Val Arg Tyr
325 330 335
Gly Ile Pro Arg Leu Asp Ile Asp Gly Phe Gly Phe Lys Pro Leu Ala
340 345 350
Asp Ser Leu Arg Lys Ile Gly Met Phe Phe Gly Ile Glu Asp Arg Ala
355 360 365
Lys Ala Ile Ile Asp Glu Glu Val Ala Arg Trp Lys Pro Glu Leu Asp
370 375 380
Trp Tyr Lys Glu Arg Leu Met Gly Lys Lys Val Cys Leu Trp Pro Gly
385 390 395 400
Gly Ser Lys Leu Trp His Trp Ala His Val Ile Glu Glu Glu Met Gly
405 410 415
Leu Lys Val Val Ser Val Tyr Thr Lys Phe Gly His Gln Gly Asp Met
420 425 430
Glu Lys Gly Ile Ala Arg Cys Gly Glu Gly Thr Leu Ala Ile Asp Asp
435 440 445
Pro Asn Glu Leu Glu Gly Leu Glu Ala Leu Glu Met Leu Lys Pro Asp
450 455 460
Ile Ile Leu Thr Gly Lys Arg Pro Gly Glu Val Ala Lys Lys Val Arg
465 470 475 480
Val Pro Tyr Leu Asn Ala His Ala Tyr His Asn Gly Pro Tyr Lys Gly
485 490 495
Phe Glu Gly Trp Val Arg Phe Ala Arg Asp Ile Tyr Asn Ala Ile Tyr
500 505 510
Ser Pro Ile His Gln Leu Ser Gly Ile Asp Ile Thr Lys Asp Asn Ala
515 520 525
Pro Glu Trp Gly Asn Gly Phe Arg Thr Arg Gln Met Leu Ser Asp Gly
530 535 540
Asn Leu Ser Asp Ala Val Arg Asn Ser Glu Thr Leu Arg Gln Tyr Thr
545 550 555 560
Gly Gly Tyr Asp Ser Val Ser Lys Leu Arg Glu Arg Glu Tyr Pro Ala
565 570 575
Phe Glu Arg Lys Val Gly Gly Gly Gly Ser Gly Gly Gly Ser Tyr Pro
580 585 590
Tyr Asp Val Pro Asp Tyr Ala Gly Gly Gly Ser Gly Gly Gly Ser Thr
595 600 605
Cys Glu Val Lys Glu Lys Gly Arg Val Gly Thr Ile Asn Pro Ile Phe
610 615 620
Thr Cys Gln Pro Ala Gly Ala Gln Phe Val Ser Ile Gly Ile Lys Asp
625 630 635 640
Cys Ile Gly Ile Val His Gly Gly Gln Gly Cys Val Met Phe Val Arg
645 650 655
Leu Ile Phe Ser Gln His Tyr Lys Glu Ser Phe Glu Leu Ala Ser Ser
660 665 670
Ser Leu His Glu Asp Gly Ala Val Phe Gly Ala Cys Gly Arg Val Glu
675 680 685
Glu Ala Val Asp Val Leu Leu Ser Arg Tyr Pro Asp Val Lys Val Val
690 695 700
Pro Ile Ile Thr Thr Cys Ser Thr Glu Ile Ile Gly Asp Asp Val Asp
705 710 715 720
Gly Val Ile Lys Lys Leu Asn Glu Gly Leu Leu Lys Glu Lys Phe Pro
725 730 735
Asp Arg Glu Val His Leu Ile Ala Met His Thr Pro Ser Phe Val Gly
740 745 750
Ser Met Ile Ser Gly Tyr Asp Val Ala Val Arg Asp Val Val Arg His
755 760 765
Phe Ala Lys Arg Glu Ala Pro Asn Asp Lys Ile Asn Leu Leu Thr Gly
770 775 780
Trp Val Asn Pro Gly Asp Val Lys Glu Leu Lys His Leu Leu Gly Glu
785 790 795 800
Met Asp Ile Glu Ala Asn Val Leu Phe Glu Ile Glu Ser Phe Asp Ser
805 810 815
Pro Ile Leu Pro Asp Gly Ser Ala Val Ser His Gly Asn Thr Thr Ile
820 825 830
Glu Asp Leu Ile Asp Thr Gly Asn Ala Arg Ala Thr Phe Ala Leu Asn
835 840 845
Arg Tyr Glu Gly Thr Lys Ala Ala Glu Tyr Leu Gln Lys Lys Phe Glu
850 855 860
Ile Pro Ala Ile Ile Gly Pro Thr Pro Ile Gly Ile Arg Asn Thr Asp
865 870 875 880
Ile Phe Leu Gln Asn Leu Lys Lys Ala Thr Gly Lys Pro Ile Pro Gln
885 890 895
Ser Leu Ala His Glu Arg Gly Val Ala Ile Asp Ala Leu Ala Asp Leu
900 905 910
Thr His Met Phe Leu Ala Glu Lys Arg Val Ala Ile Tyr Gly Ala Pro
915 920 925
Asp Leu Val Ile Gly Leu Ala Glu Phe Cys Leu Asp Leu Glu Met Lys
930 935 940
Pro Val Leu Leu Leu Leu Gly Asp Asp Asn Ser Lys Tyr Val Asp Asp
945 950 955 960
Pro Arg Ile Lys Ala Leu Gln Glu Asn Val Asp Tyr Gly Met Glu Ile
965 970 975
Val Thr Asn Ala Asp Phe Trp Glu Leu Glu Asn Arg Ile Lys Asn Glu
980 985 990
Gly Leu Glu Leu Asp Leu Ile Leu Gly His Ser Lys Gly Arg Phe Ile
995 1000 1005
Ser Ile Asp Tyr Asn Ile Pro Met Leu Arg Val Gly Phe Pro Thr
1010 1015 1020
Tyr Asp Arg Ala Gly Leu Phe Arg Tyr Pro Thr Val Gly Tyr Gly
1025 1030 1035
Gly Ala Ile Trp Leu Ala Glu Gln Met Ala Asn Thr Leu Phe Ala
1040 1045 1050
Asp Met Glu His Lys Lys Asn Lys Glu Trp Val Leu Asn Val Trp
1055 1060 1065
<210> 203
<211> 1065
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность пептида MTP-
CoxIV::AnfD::линкер26(HA)::AnfK, кодированная SN273. Аминокислоты 1-61
соответствуют последовательности MTP-CoxIV, включая GG на ее C-конце,
аминокислоты 62-578 соответствуют последовательности AnfD
(A. vinelandii),
<400> 203
Met Leu Ser Leu Arg Gln Ser Ile Arg Phe Phe Lys Pro Ala Thr Arg
1. 5 10 15
Thr Leu Cys Ser Ser Arg Tyr Leu Leu Gln Gln Lys Pro Ser Ala Trp
20 25 30
Ser His Pro Gln Phe Glu Lys Gly Gly Gly Ser Gly Gly Gly Ser Gly
35 40 45
Gly Ser Ala Trp Ser His Pro Gln Phe Glu Lys Gly Gly Met Pro His
50 55 60
His Glu Phe Glu Cys Ser Lys Val Ile Pro Glu Arg Lys Lys His Ala
65 70 75 80
Val Ile Lys Gly Lys Gly Glu Thr Leu Ala Asp Ala Leu Pro Gln Gly
85 90 95
Tyr Leu Asn Thr Ile Pro Gly Ser Ile Ser Glu Arg Gly Cys Ala Tyr
100 105 110
Cys Gly Ala Lys His Val Ile Gly Thr Pro Met Lys Asp Val Ile His
115 120 125
Ile Ser His Gly Pro Val Gly Cys Thr Tyr Asp Thr Trp Gln Thr Lys
130 135 140
Arg Tyr Ile Ser Asp Asn Asp Asn Phe Gln Leu Lys Tyr Thr Tyr Ala
145 150 155 160
Thr Asp Val Lys Glu Lys His Ile Val Phe Gly Ala Glu Lys Leu Leu
165 170 175
Lys Gln Asn Ile Ile Glu Ala Phe Lys Ala Phe Pro Gln Ile Lys Arg
180 185 190
Met Thr Ile Tyr Gln Thr Cys Ala Thr Ala Leu Ile Gly Asp Asp Ile
195 200 205
Asn Ala Ile Ala Glu Glu Val Met Glu Glu Met Pro Glu Val Asp Ile
210 215 220
Phe Val Cys Asn Ser Pro Gly Phe Ala Gly Pro Ser Gln Ser Gly Gly
225 230 235 240
His His Lys Ile Asn Ile Ala Trp Ile Asn Gln Lys Val Gly Thr Val
245 250 255
Glu Pro Glu Ile Thr Gly Asp His Val Ile Asn Tyr Val Gly Glu Tyr
260 265 270
Asn Ile Gln Gly Asp Gln Glu Val Met Val Asp Tyr Phe Lys Arg Met
275 280 285
Gly Ile Gln Val Leu Ser Thr Phe Thr Gly Asn Gly Ser Tyr Asp Gly
290 295 300
Leu Arg Ala Met His Arg Ala His Leu Asn Val Leu Glu Cys Ala Arg
305 310 315 320
Ser Ala Glu Tyr Ile Cys Asn Glu Leu Arg Val Arg Tyr Gly Ile Pro
325 330 335
Arg Leu Asp Ile Asp Gly Phe Gly Phe Lys Pro Leu Ala Asp Ser Leu
340 345 350
Arg Lys Ile Gly Met Phe Phe Gly Ile Glu Asp Arg Ala Lys Ala Ile
355 360 365
Ile Asp Glu Glu Val Ala Arg Trp Lys Pro Glu Leu Asp Trp Tyr Lys
370 375 380
Glu Arg Leu Met Gly Lys Lys Val Cys Leu Trp Pro Gly Gly Ser Lys
385 390 395 400
Leu Trp His Trp Ala His Val Ile Glu Glu Glu Met Gly Leu Lys Val
405 410 415
Val Ser Val Tyr Thr Lys Phe Gly His Gln Gly Asp Met Glu Lys Gly
420 425 430
Ile Ala Arg Cys Gly Glu Gly Thr Leu Ala Ile Asp Asp Pro Asn Glu
435 440 445
Leu Glu Gly Leu Glu Ala Leu Glu Met Leu Lys Pro Asp Ile Ile Leu
450 455 460
Thr Gly Lys Arg Pro Gly Glu Val Ala Lys Lys Val Arg Val Pro Tyr
465 470 475 480
Leu Asn Ala His Ala Tyr His Asn Gly Pro Tyr Lys Gly Phe Glu Gly
485 490 495
Trp Val Arg Phe Ala Arg Asp Ile Tyr Asn Ala Ile Tyr Ser Pro Ile
500 505 510
His Gln Leu Ser Gly Ile Asp Ile Thr Lys Asp Asn Ala Pro Glu Trp
515 520 525
Gly Asn Gly Phe Arg Thr Arg Gln Met Leu Ser Asp Gly Asn Leu Ser
530 535 540
Asp Ala Val Arg Asn Ser Glu Thr Leu Arg Gln Tyr Thr Gly Gly Tyr
545 550 555 560
Asp Ser Val Ser Lys Leu Arg Glu Arg Glu Tyr Pro Ala Phe Glu Arg
565 570 575
Lys Val Gly Gly Gly Gly Ser Gly Gly Gly Ser Tyr Pro Tyr Asp Val
580 585 590
Pro Asp Tyr Ala Gly Gly Gly Ser Gly Gly Gly Ser Thr Cys Glu Val
595 600 605
Lys Glu Lys Gly Arg Val Gly Thr Ile Asn Pro Ile Phe Thr Cys Gln
610 615 620
Pro Ala Gly Ala Gln Phe Val Ser Ile Gly Ile Lys Asp Cys Ile Gly
625 630 635 640
Ile Val His Gly Gly Gln Gly Cys Val Met Phe Val Arg Leu Ile Phe
645 650 655
Ser Gln His Tyr Lys Glu Ser Phe Glu Leu Ala Ser Ser Ser Leu His
660 665 670
Glu Asp Gly Ala Val Phe Gly Ala Cys Gly Arg Val Glu Glu Ala Val
675 680 685
Asp Val Leu Leu Ser Arg Tyr Pro Asp Val Lys Val Val Pro Ile Ile
690 695 700
Thr Thr Cys Ser Thr Glu Ile Ile Gly Asp Asp Val Asp Gly Val Ile
705 710 715 720
Lys Lys Leu Asn Glu Gly Leu Leu Lys Glu Lys Phe Pro Asp Arg Glu
725 730 735
Val His Leu Ile Ala Met His Thr Pro Ser Phe Val Gly Ser Met Ile
740 745 750
Ser Gly Tyr Asp Val Ala Val Arg Asp Val Val Arg His Phe Ala Lys
755 760 765
Arg Glu Ala Pro Asn Asp Lys Ile Asn Leu Leu Thr Gly Trp Val Asn
770 775 780
Pro Gly Asp Val Lys Glu Leu Lys His Leu Leu Gly Glu Met Asp Ile
785 790 795 800
Glu Ala Asn Val Leu Phe Glu Ile Glu Ser Phe Asp Ser Pro Ile Leu
805 810 815
Pro Asp Gly Ser Ala Val Ser His Gly Asn Thr Thr Ile Glu Asp Leu
820 825 830
Ile Asp Thr Gly Asn Ala Arg Ala Thr Phe Ala Leu Asn Arg Tyr Glu
835 840 845
Gly Thr Lys Ala Ala Glu Tyr Leu Gln Lys Lys Phe Glu Ile Pro Ala
850 855 860
Ile Ile Gly Pro Thr Pro Ile Gly Ile Arg Asn Thr Asp Ile Phe Leu
865 870 875 880
Gln Asn Leu Lys Lys Ala Thr Gly Lys Pro Ile Pro Gln Ser Leu Ala
885 890 895
His Glu Arg Gly Val Ala Ile Asp Ala Leu Ala Asp Leu Thr His Met
900 905 910
Phe Leu Ala Glu Lys Arg Val Ala Ile Tyr Gly Ala Pro Asp Leu Val
915 920 925
Ile Gly Leu Ala Glu Phe Cys Leu Asp Leu Glu Met Lys Pro Val Leu
930 935 940
Leu Leu Leu Gly Asp Asp Asn Ser Lys Tyr Val Asp Asp Pro Arg Ile
945 950 955 960
Lys Ala Leu Gln Glu Asn Val Asp Tyr Gly Met Glu Ile Val Thr Asn
965 970 975
Ala Asp Phe Trp Glu Leu Glu Asn Arg Ile Lys Asn Glu Gly Leu Glu
980 985 990
Leu Asp Leu Ile Leu Gly His Ser Lys Gly Arg Phe Ile Ser Ile Asp
995 1000 1005
Tyr Asn Ile Pro Met Leu Arg Val Gly Phe Pro Thr Tyr Asp Arg
1010 1015 1020
Ala Gly Leu Phe Arg Tyr Pro Thr Val Gly Tyr Gly Gly Ala Ile
1025 1030 1035
Trp Leu Ala Glu Gln Met Ala Asn Thr Leu Phe Ala Asp Met Glu
1040 1045 1050
His Lys Lys Asn Lys Glu Trp Val Leu Asn Val Trp
1055 1060 1065
<210> 204
<211> 1068
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность пептида
mFA?51::AnfD::линкер26(HA)::AnfK, кодированная SN274. Аминокислоты
1-64 соответствуют последовательности mFA?51, включая замену аланина,
которая не допускает MPP-расщепление, и включая GG на его C-конце,
аминокислоты
<400> 204
Met Ala Met Ala Val Phe Arg Arg Glu Ala Ala Ala Leu Leu Pro Ser
1. 5 10 15
Ile Ala Ala Arg Pro Ile Ala Ala Ala Ala Ala Ala Ala Ser Ser Asp
20 25 30
Gln Glu Glu Gly Leu Leu Ala Ala Ala Ala Ala Ala Ala Ala Val Val
35 40 45
Arg Asn Arg Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Gly Gly
50 55 60
Met Pro His His Glu Phe Glu Cys Ser Lys Val Ile Pro Glu Arg Lys
65 70 75 80
Lys His Ala Val Ile Lys Gly Lys Gly Glu Thr Leu Ala Asp Ala Leu
85 90 95
Pro Gln Gly Tyr Leu Asn Thr Ile Pro Gly Ser Ile Ser Glu Arg Gly
100 105 110
Cys Ala Tyr Cys Gly Ala Lys His Val Ile Gly Thr Pro Met Lys Asp
115 120 125
Val Ile His Ile Ser His Gly Pro Val Gly Cys Thr Tyr Asp Thr Trp
130 135 140
Gln Thr Lys Arg Tyr Ile Ser Asp Asn Asp Asn Phe Gln Leu Lys Tyr
145 150 155 160
Thr Tyr Ala Thr Asp Val Lys Glu Lys His Ile Val Phe Gly Ala Glu
165 170 175
Lys Leu Leu Lys Gln Asn Ile Ile Glu Ala Phe Lys Ala Phe Pro Gln
180 185 190
Ile Lys Arg Met Thr Ile Tyr Gln Thr Cys Ala Thr Ala Leu Ile Gly
195 200 205
Asp Asp Ile Asn Ala Ile Ala Glu Glu Val Met Glu Glu Met Pro Glu
210 215 220
Val Asp Ile Phe Val Cys Asn Ser Pro Gly Phe Ala Gly Pro Ser Gln
225 230 235 240
Ser Gly Gly His His Lys Ile Asn Ile Ala Trp Ile Asn Gln Lys Val
245 250 255
Gly Thr Val Glu Pro Glu Ile Thr Gly Asp His Val Ile Asn Tyr Val
260 265 270
Gly Glu Tyr Asn Ile Gln Gly Asp Gln Glu Val Met Val Asp Tyr Phe
275 280 285
Lys Arg Met Gly Ile Gln Val Leu Ser Thr Phe Thr Gly Asn Gly Ser
290 295 300
Tyr Asp Gly Leu Arg Ala Met His Arg Ala His Leu Asn Val Leu Glu
305 310 315 320
Cys Ala Arg Ser Ala Glu Tyr Ile Cys Asn Glu Leu Arg Val Arg Tyr
325 330 335
Gly Ile Pro Arg Leu Asp Ile Asp Gly Phe Gly Phe Lys Pro Leu Ala
340 345 350
Asp Ser Leu Arg Lys Ile Gly Met Phe Phe Gly Ile Glu Asp Arg Ala
355 360 365
Lys Ala Ile Ile Asp Glu Glu Val Ala Arg Trp Lys Pro Glu Leu Asp
370 375 380
Trp Tyr Lys Glu Arg Leu Met Gly Lys Lys Val Cys Leu Trp Pro Gly
385 390 395 400
Gly Ser Lys Leu Trp His Trp Ala His Val Ile Glu Glu Glu Met Gly
405 410 415
Leu Lys Val Val Ser Val Tyr Thr Lys Phe Gly His Gln Gly Asp Met
420 425 430
Glu Lys Gly Ile Ala Arg Cys Gly Glu Gly Thr Leu Ala Ile Asp Asp
435 440 445
Pro Asn Glu Leu Glu Gly Leu Glu Ala Leu Glu Met Leu Lys Pro Asp
450 455 460
Ile Ile Leu Thr Gly Lys Arg Pro Gly Glu Val Ala Lys Lys Val Arg
465 470 475 480
Val Pro Tyr Leu Asn Ala His Ala Tyr His Asn Gly Pro Tyr Lys Gly
485 490 495
Phe Glu Gly Trp Val Arg Phe Ala Arg Asp Ile Tyr Asn Ala Ile Tyr
500 505 510
Ser Pro Ile His Gln Leu Ser Gly Ile Asp Ile Thr Lys Asp Asn Ala
515 520 525
Pro Glu Trp Gly Asn Gly Phe Arg Thr Arg Gln Met Leu Ser Asp Gly
530 535 540
Asn Leu Ser Asp Ala Val Arg Asn Ser Glu Thr Leu Arg Gln Tyr Thr
545 550 555 560
Gly Gly Tyr Asp Ser Val Ser Lys Leu Arg Glu Arg Glu Tyr Pro Ala
565 570 575
Phe Glu Arg Lys Val Gly Gly Gly Gly Ser Gly Gly Gly Ser Tyr Pro
580 585 590
Tyr Asp Val Pro Asp Tyr Ala Gly Gly Gly Ser Gly Gly Gly Ser Thr
595 600 605
Cys Glu Val Lys Glu Lys Gly Arg Val Gly Thr Ile Asn Pro Ile Phe
610 615 620
Thr Cys Gln Pro Ala Gly Ala Gln Phe Val Ser Ile Gly Ile Lys Asp
625 630 635 640
Cys Ile Gly Ile Val His Gly Gly Gln Gly Cys Val Met Phe Val Arg
645 650 655
Leu Ile Phe Ser Gln His Tyr Lys Glu Ser Phe Glu Leu Ala Ser Ser
660 665 670
Ser Leu His Glu Asp Gly Ala Val Phe Gly Ala Cys Gly Arg Val Glu
675 680 685
Glu Ala Val Asp Val Leu Leu Ser Arg Tyr Pro Asp Val Lys Val Val
690 695 700
Pro Ile Ile Thr Thr Cys Ser Thr Glu Ile Ile Gly Asp Asp Val Asp
705 710 715 720
Gly Val Ile Lys Lys Leu Asn Glu Gly Leu Leu Lys Glu Lys Phe Pro
725 730 735
Asp Arg Glu Val His Leu Ile Ala Met His Thr Pro Ser Phe Val Gly
740 745 750
Ser Met Ile Ser Gly Tyr Asp Val Ala Val Arg Asp Val Val Arg His
755 760 765
Phe Ala Lys Arg Glu Ala Pro Asn Asp Lys Ile Asn Leu Leu Thr Gly
770 775 780
Trp Val Asn Pro Gly Asp Val Lys Glu Leu Lys His Leu Leu Gly Glu
785 790 795 800
Met Asp Ile Glu Ala Asn Val Leu Phe Glu Ile Glu Ser Phe Asp Ser
805 810 815
Pro Ile Leu Pro Asp Gly Ser Ala Val Ser His Gly Asn Thr Thr Ile
820 825 830
Glu Asp Leu Ile Asp Thr Gly Asn Ala Arg Ala Thr Phe Ala Leu Asn
835 840 845
Arg Tyr Glu Gly Thr Lys Ala Ala Glu Tyr Leu Gln Lys Lys Phe Glu
850 855 860
Ile Pro Ala Ile Ile Gly Pro Thr Pro Ile Gly Ile Arg Asn Thr Asp
865 870 875 880
Ile Phe Leu Gln Asn Leu Lys Lys Ala Thr Gly Lys Pro Ile Pro Gln
885 890 895
Ser Leu Ala His Glu Arg Gly Val Ala Ile Asp Ala Leu Ala Asp Leu
900 905 910
Thr His Met Phe Leu Ala Glu Lys Arg Val Ala Ile Tyr Gly Ala Pro
915 920 925
Asp Leu Val Ile Gly Leu Ala Glu Phe Cys Leu Asp Leu Glu Met Lys
930 935 940
Pro Val Leu Leu Leu Leu Gly Asp Asp Asn Ser Lys Tyr Val Asp Asp
945 950 955 960
Pro Arg Ile Lys Ala Leu Gln Glu Asn Val Asp Tyr Gly Met Glu Ile
965 970 975
Val Thr Asn Ala Asp Phe Trp Glu Leu Glu Asn Arg Ile Lys Asn Glu
980 985 990
Gly Leu Glu Leu Asp Leu Ile Leu Gly His Ser Lys Gly Arg Phe Ile
995 1000 1005
Ser Ile Asp Tyr Asn Ile Pro Met Leu Arg Val Gly Phe Pro Thr
1010 1015 1020
Tyr Asp Arg Ala Gly Leu Phe Arg Tyr Pro Thr Val Gly Tyr Gly
1025 1030 1035
Gly Ala Ile Trp Leu Ala Glu Gln Met Ala Asn Thr Leu Phe Ala
1040 1045 1050
Asp Met Glu His Lys Lys Asn Lys Glu Trp Val Leu Asn Val Trp
1055 1060 1065
<210> 205
<211> 1013
<212> PRT
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность полипептида
HISx6::AnfD::линкер26(HA)::AnfK, кодированная SN275, которая не имеет
последовательности MTP и может быть локализована в цитоплазме.
Аминокислоты 1-9 соответствуют последовательности HISx6, включая GG на
ее C-конце, амино
<400> 205
Met His His His His His His Gly Gly Met Pro His His Glu Phe Glu
1. 5 10 15
Cys Ser Lys Val Ile Pro Glu Arg Lys Lys His Ala Val Ile Lys Gly
20 25 30
Lys Gly Glu Thr Leu Ala Asp Ala Leu Pro Gln Gly Tyr Leu Asn Thr
35 40 45
Ile Pro Gly Ser Ile Ser Glu Arg Gly Cys Ala Tyr Cys Gly Ala Lys
50 55 60
His Val Ile Gly Thr Pro Met Lys Asp Val Ile His Ile Ser His Gly
65 70 75 80
Pro Val Gly Cys Thr Tyr Asp Thr Trp Gln Thr Lys Arg Tyr Ile Ser
85 90 95
Asp Asn Asp Asn Phe Gln Leu Lys Tyr Thr Tyr Ala Thr Asp Val Lys
100 105 110
Glu Lys His Ile Val Phe Gly Ala Glu Lys Leu Leu Lys Gln Asn Ile
115 120 125
Ile Glu Ala Phe Lys Ala Phe Pro Gln Ile Lys Arg Met Thr Ile Tyr
130 135 140
Gln Thr Cys Ala Thr Ala Leu Ile Gly Asp Asp Ile Asn Ala Ile Ala
145 150 155 160
Glu Glu Val Met Glu Glu Met Pro Glu Val Asp Ile Phe Val Cys Asn
165 170 175
Ser Pro Gly Phe Ala Gly Pro Ser Gln Ser Gly Gly His His Lys Ile
180 185 190
Asn Ile Ala Trp Ile Asn Gln Lys Val Gly Thr Val Glu Pro Glu Ile
195 200 205
Thr Gly Asp His Val Ile Asn Tyr Val Gly Glu Tyr Asn Ile Gln Gly
210 215 220
Asp Gln Glu Val Met Val Asp Tyr Phe Lys Arg Met Gly Ile Gln Val
225 230 235 240
Leu Ser Thr Phe Thr Gly Asn Gly Ser Tyr Asp Gly Leu Arg Ala Met
245 250 255
His Arg Ala His Leu Asn Val Leu Glu Cys Ala Arg Ser Ala Glu Tyr
260 265 270
Ile Cys Asn Glu Leu Arg Val Arg Tyr Gly Ile Pro Arg Leu Asp Ile
275 280 285
Asp Gly Phe Gly Phe Lys Pro Leu Ala Asp Ser Leu Arg Lys Ile Gly
290 295 300
Met Phe Phe Gly Ile Glu Asp Arg Ala Lys Ala Ile Ile Asp Glu Glu
305 310 315 320
Val Ala Arg Trp Lys Pro Glu Leu Asp Trp Tyr Lys Glu Arg Leu Met
325 330 335
Gly Lys Lys Val Cys Leu Trp Pro Gly Gly Ser Lys Leu Trp His Trp
340 345 350
Ala His Val Ile Glu Glu Glu Met Gly Leu Lys Val Val Ser Val Tyr
355 360 365
Thr Lys Phe Gly His Gln Gly Asp Met Glu Lys Gly Ile Ala Arg Cys
370 375 380
Gly Glu Gly Thr Leu Ala Ile Asp Asp Pro Asn Glu Leu Glu Gly Leu
385 390 395 400
Glu Ala Leu Glu Met Leu Lys Pro Asp Ile Ile Leu Thr Gly Lys Arg
405 410 415
Pro Gly Glu Val Ala Lys Lys Val Arg Val Pro Tyr Leu Asn Ala His
420 425 430
Ala Tyr His Asn Gly Pro Tyr Lys Gly Phe Glu Gly Trp Val Arg Phe
435 440 445
Ala Arg Asp Ile Tyr Asn Ala Ile Tyr Ser Pro Ile His Gln Leu Ser
450 455 460
Gly Ile Asp Ile Thr Lys Asp Asn Ala Pro Glu Trp Gly Asn Gly Phe
465 470 475 480
Arg Thr Arg Gln Met Leu Ser Asp Gly Asn Leu Ser Asp Ala Val Arg
485 490 495
Asn Ser Glu Thr Leu Arg Gln Tyr Thr Gly Gly Tyr Asp Ser Val Ser
500 505 510
Lys Leu Arg Glu Arg Glu Tyr Pro Ala Phe Glu Arg Lys Val Gly Gly
515 520 525
Gly Gly Ser Gly Gly Gly Ser Tyr Pro Tyr Asp Val Pro Asp Tyr Ala
530 535 540
Gly Gly Gly Ser Gly Gly Gly Ser Thr Cys Glu Val Lys Glu Lys Gly
545 550 555 560
Arg Val Gly Thr Ile Asn Pro Ile Phe Thr Cys Gln Pro Ala Gly Ala
565 570 575
Gln Phe Val Ser Ile Gly Ile Lys Asp Cys Ile Gly Ile Val His Gly
580 585 590
Gly Gln Gly Cys Val Met Phe Val Arg Leu Ile Phe Ser Gln His Tyr
595 600 605
Lys Glu Ser Phe Glu Leu Ala Ser Ser Ser Leu His Glu Asp Gly Ala
610 615 620
Val Phe Gly Ala Cys Gly Arg Val Glu Glu Ala Val Asp Val Leu Leu
625 630 635 640
Ser Arg Tyr Pro Asp Val Lys Val Val Pro Ile Ile Thr Thr Cys Ser
645 650 655
Thr Glu Ile Ile Gly Asp Asp Val Asp Gly Val Ile Lys Lys Leu Asn
660 665 670
Glu Gly Leu Leu Lys Glu Lys Phe Pro Asp Arg Glu Val His Leu Ile
675 680 685
Ala Met His Thr Pro Ser Phe Val Gly Ser Met Ile Ser Gly Tyr Asp
690 695 700
Val Ala Val Arg Asp Val Val Arg His Phe Ala Lys Arg Glu Ala Pro
705 710 715 720
Asn Asp Lys Ile Asn Leu Leu Thr Gly Trp Val Asn Pro Gly Asp Val
725 730 735
Lys Glu Leu Lys His Leu Leu Gly Glu Met Asp Ile Glu Ala Asn Val
740 745 750
Leu Phe Glu Ile Glu Ser Phe Asp Ser Pro Ile Leu Pro Asp Gly Ser
755 760 765
Ala Val Ser His Gly Asn Thr Thr Ile Glu Asp Leu Ile Asp Thr Gly
770 775 780
Asn Ala Arg Ala Thr Phe Ala Leu Asn Arg Tyr Glu Gly Thr Lys Ala
785 790 795 800
Ala Glu Tyr Leu Gln Lys Lys Phe Glu Ile Pro Ala Ile Ile Gly Pro
805 810 815
Thr Pro Ile Gly Ile Arg Asn Thr Asp Ile Phe Leu Gln Asn Leu Lys
820 825 830
Lys Ala Thr Gly Lys Pro Ile Pro Gln Ser Leu Ala His Glu Arg Gly
835 840 845
Val Ala Ile Asp Ala Leu Ala Asp Leu Thr His Met Phe Leu Ala Glu
850 855 860
Lys Arg Val Ala Ile Tyr Gly Ala Pro Asp Leu Val Ile Gly Leu Ala
865 870 875 880
Glu Phe Cys Leu Asp Leu Glu Met Lys Pro Val Leu Leu Leu Leu Gly
885 890 895
Asp Asp Asn Ser Lys Tyr Val Asp Asp Pro Arg Ile Lys Ala Leu Gln
900 905 910
Glu Asn Val Asp Tyr Gly Met Glu Ile Val Thr Asn Ala Asp Phe Trp
915 920 925
Glu Leu Glu Asn Arg Ile Lys Asn Glu Gly Leu Glu Leu Asp Leu Ile
930 935 940
Leu Gly His Ser Lys Gly Arg Phe Ile Ser Ile Asp Tyr Asn Ile Pro
945 950 955 960
Met Leu Arg Val Gly Phe Pro Thr Tyr Asp Arg Ala Gly Leu Phe Arg
965 970 975
Tyr Pro Thr Val Gly Tyr Gly Gly Ala Ile Trp Leu Ala Glu Gln Met
980 985 990
Ala Asn Thr Leu Phe Ala Asp Met Glu His Lys Lys Asn Lys Glu Trp
995 1000 1005
Val Leu Asn Val Trp
1010
<210> 206
<211> 383
<212> PRT
<213> Thermoanaerobacter brockii
<400> 206
Met Thr Leu Lys Lys Gly Lys Lys Val Tyr Ile Val Asp Thr Thr Leu
1. 5 10 15
Arg Asp Gly Glu Gln Thr Ala Gly Val Val Phe Ala Asn Asn Glu Lys
20 25 30
Ile Arg Ile Ala Gln Met Leu Asp Glu Ile Gly Ile Asp Gln Leu Glu
35 40 45
Val Gly Ile Pro Thr Met Gly Gly Asp Glu Lys Glu Thr Val Thr Lys
50 55 60
Ile Ala Lys Leu Gly Leu Asn Ala Ser Ile Met Ala Trp Asn Arg Ala
65 70 75 80
Val Val Lys Asp Val Gln Glu Ser Leu Glu Cys Gly Val Asp Ala Val
85 90 95
Ala Ile Ser Val Ser Thr Ser Asp Ile His Ile Glu His Lys Leu Lys
100 105 110
Lys Thr Arg Gln Trp Val Leu Asp Asn Met Thr Glu Ala Val Lys Phe
115 120 125
Ala Lys Lys Glu Gly Val Tyr Val Ser Val Asn Ala Glu Asp Ala Ser
130 135 140
Arg Thr Asp Met Asn Phe Leu Ile Glu Phe Ala Lys Cys Ala Lys Gln
145 150 155 160
Ala Gly Ala Asp Arg Leu Arg Phe Cys Asp Thr Val Gly Phe Leu Asp
165 170 175
Pro Phe Lys Thr Tyr Asp Met Val Lys Ala Ile Lys Glu Ala Val Asp
180 185 190
Ile Asp Ile Glu Met His Thr His Asn Asp Phe Gly Met Ala Thr Ala
195 200 205
Asn Ala Leu Ala Gly Met Arg Ala Gly Ala Asn Phe Ile Gly Val Thr
210 215 220
Val Asn Gly Leu Gly Glu Arg Ala Gly Asn Ala Ala Leu Glu Glu Val
225 230 235 240
Val Met Ala Leu Lys His Val Tyr Lys Ile Asp Leu Gly Ile Asp Thr
245 250 255
Thr Arg Phe Arg Glu Ile Ser Glu Tyr Val Ala Leu Ala Ser Gly Arg
260 265 270
Gln Leu Pro Ala Trp Lys Ala Ile Val Gly Thr Asn Val Phe Ala His
275 280 285
Glu Ser Gly Ile His Val Asp Gly Ala Leu Lys Asn Pro His Thr Tyr
290 295 300
Glu Ile Phe Asn Pro Asp Glu Val Gly Leu Glu Arg Gln Ile Val Ile
305 310 315 320
Gly Lys His Ser Gly Thr Ala Ala Leu Ile Asn Lys Phe Lys Glu Tyr
325 330 335
Gly Arg Val Leu Thr Glu Glu Glu Ala Asn Leu Leu Leu Pro His Val
340 345 350
Arg Lys Leu Ala Ile Gln Leu Lys Arg Pro Leu Phe Asp Lys Glu Leu
355 360 365
Met Tyr Leu Tyr Glu Asp Val Ile Lys Asn Arg Glu Lys Ala Ile
370 375 380
<210> 207
<211> 378
<212> PRT
<213> Thermincola potens
<400> 207
Met Met Glu Lys Lys Ile Thr Ile Val Asp Thr Thr Leu Arg Asp Gly
1. 5 10 15
Glu Gln Thr Ala Gly Val Val Phe Ala Asn Lys Glu Lys Val Arg Ile
20 25 30
Ala Gln Met Leu Asp Glu Leu Gly Val His Gln Ile Glu Ala Gly Ile
35 40 45
Pro Val Met Gly Gly Asp Glu Glu Lys Ala Val Lys Asp Ile Val Lys
50 55 60
Leu Gly Leu Lys Ala Ser Ile Met Gly Trp Asn Arg Ala Val Ile Ser
65 70 75 80
Asp Ile Glu Glu Thr Leu Arg Cys Gly Val Asp Ala Val Ala Ile Ser
85 90 95
Ile Ser Thr Ser Asp Ile His Ile Gln His Lys Leu Gln Thr Ser Arg
100 105 110
Glu Trp Val Leu Glu Asn Met Val Lys Ala Val Glu Phe Ala Lys Lys
115 120 125
Glu Gly Val Tyr Ile Ser Val Asn Ala Glu Asp Ala Ser Arg Ser Asp
130 135 140
Met Glu Phe Leu Ile Gln Phe Ala Arg Ala Ala Lys Glu Ala Gly Ala
145 150 155 160
Asp Arg Ile Arg Tyr Cys Asp Thr Ile Gly Ile Leu Asp Pro Phe Thr
165 170 175
Thr Tyr Glu Asn Ile Gln Thr Leu Lys Lys His Val Asp Ile Asp Ile
180 185 190
Glu Met His Thr His Asn Asp Phe Gly Met Ala Thr Ala Asn Ala Leu
195 200 205
Ala Gly Ile Lys Ala Gly Ala Ser His Val Gly Val Thr Val Met Gly
210 215 220
Leu Gly Glu Arg Ala Gly Asn Ala Ala Leu Glu Glu Val Val Met Ala
225 230 235 240
Leu Lys His Ile Trp Gln Ile Asp Leu Gly Phe Lys Thr Asn Met Phe
245 250 255
Arg Asp Leu Ala Glu Tyr Val Ser Leu Ala Ser Gly Arg Glu Leu Pro
260 265 270
Ala Trp Lys Ala Ile Val Gly Ser Asn Met Phe Ala His Glu Ser Gly
275 280 285
Ile His Ala Asp Gly Ala Ile Lys Asn Pro Ile Thr Tyr Glu Val Phe
290 295 300
Ala Pro Glu Glu Val Gly Leu Glu Arg Gln Ile Val Ile Gly Lys His
305 310 315 320
Ser Gly Ser Lys Ala Leu Met Met Lys Phe Ala Glu Tyr Gly Ile His
325 330 335
Leu Ser Glu Ala Asp Ala Ala Gln Leu Leu Pro Lys Ile Arg Ser His
340 345 350
Ala Val Ala Leu Lys Arg Ser Leu Phe Asp Lys Glu Leu Val Tyr Ile
355 360 365
Tyr Glu Glu Val Phe Gly Lys Lys Pro Leu
370 375
<210> 208
<211> 440
<212> PRT
<213> Saccharomyces cerevisiae
<400> 208
Met Ser Glu Asn Asn Glu Phe Gln Ser Val Thr Glu Ser Thr Thr Ala
1. 5 10 15
Pro Thr Thr Ser Asn Pro Tyr Gly Pro Asn Pro Ala Asp Tyr Leu Ser
20 25 30
Asn Val Lys Asn Phe Gln Leu Ile Asp Ser Thr Leu Arg Glu Gly Glu
35 40 45
Gln Phe Ala Asn Ala Phe Phe Asp Thr Glu Lys Lys Ile Glu Ile Ala
50 55 60
Arg Ala Leu Asp Asp Phe Gly Val Asp Tyr Ile Glu Leu Thr Ser Pro
65 70 75 80
Val Ala Ser Glu Gln Ser Arg Lys Asp Cys Glu Ala Ile Cys Lys Leu
85 90 95
Gly Leu Lys Ala Lys Ile Leu Thr His Ile Arg Cys His Met Asp Asp
100 105 110
Ala Arg Val Ala Val Glu Thr Gly Val Asp Gly Val Asp Val Val Ile
115 120 125
Gly Thr Ser Lys Phe Leu Arg Gln Tyr Ser His Gly Lys Asp Met Asn
130 135 140
Tyr Ile Ala Lys Ser Ala Val Glu Val Ile Glu Phe Val Lys Ser Lys
145 150 155 160
Gly Ile Glu Ile Arg Phe Ser Ser Glu Asp Ser Phe Arg Ser Asp Leu
165 170 175
Val Asp Leu Leu Asn Ile Tyr Lys Thr Val Asp Lys Ile Gly Val Asn
180 185 190
Arg Val Gly Ile Ala Asp Thr Val Gly Cys Ala Asn Pro Arg Gln Val
195 200 205
Tyr Glu Leu Ile Arg Thr Leu Lys Ser Val Val Ser Cys Asp Ile Glu
210 215 220
Cys His Phe His Asn Asp Thr Gly Cys Ala Ile Ala Asn Ala Tyr Thr
225 230 235 240
Ala Leu Glu Gly Gly Ala Arg Leu Ile Asp Val Ser Val Leu Gly Ile
245 250 255
Gly Glu Arg Asn Gly Ile Thr Pro Leu Gly Gly Leu Met Ala Arg Met
260 265 270
Ile Val Ala Ala Pro Asp Tyr Val Arg Ser Lys Tyr Lys Leu His Lys
275 280 285
Ile Arg Asp Ile Glu Asn Leu Val Ala Asp Ala Val Glu Val Asn Ile
290 295 300
Pro Phe Asn Asn Pro Ile Thr Gly Phe Cys Ala Phe Thr His Lys Ala
305 310 315 320
Gly Ile His Ala Lys Ala Ile Leu Ala Asn Pro Ser Thr Tyr Glu Ile
325 330 335
Leu Asp Pro His Asp Phe Gly Met Lys Arg Tyr Ile His Phe Ala Asn
340 345 350
Arg Leu Thr Gly Trp Asn Ala Ile Lys Ser Arg Val Asp Gln Leu Asn
355 360 365
Leu Asn Leu Thr Asp Asp Gln Ile Lys Glu Val Thr Ala Lys Ile Lys
370 375 380
Lys Leu Gly Asp Val Arg Pro Leu Asn Ile Asp Asp Val Asp Ser Ile
385 390 395 400
Ile Lys Asp Phe His Ala Glu Leu Ser Thr Pro Leu Leu Lys Pro Val
405 410 415
Asn Lys Gly Thr Asp Asp Asp Asn Ile Asp Ile Ser Asn Gly His Val
420 425 430
Ser Lys Lys Ala Lys Val Thr Lys
435 440
<210> 209
<211> 376
<212> PRT
<213> Nodularia spumigena
<400> 209
Met Asn Glu Ile Leu Ile Asn Asp Thr Thr Leu Arg Asp Gly Glu Gln
1. 5 10 15
Ala Ala Gly Val Ala Phe Thr Phe Glu Glu Lys Val Ala Ile Ala Gln
20 25 30
Phe Leu Asp Ala Ile Gly Val Pro Glu Leu Glu Val Gly Ile Pro Ala
35 40 45
Met Gly Glu Ala Glu Thr His Ala Ile Leu Ala Ile Ser Asp Leu Gly
50 55 60
Leu Gln Ala Ser Leu Leu Gly Trp Asn Arg Ala Val Leu Ser Asp Ile
65 70 75 80
Lys Ala Ser Ile Thr Cys Gly Leu Lys Arg Val His Ile Ala Ile Pro
85 90 95
Val Ser Gly Ile Gln Ile Ala Ala Lys Phe His Gly Gln Trp Arg Val
100 105 110
Ser Leu Gln Arg Leu Lys Asp Cys Ile Ser Phe Ala Val Asp Gln Gly
115 120 125
Leu Trp Val Ala Val Gly Gly Glu Asp Ser Ser Arg Ala Asp Pro Asn
130 135 140
Phe Leu Leu Asp Val Ala Leu Asn Ala Gln Glu Trp Gly Ala Ser Arg
145 150 155 160
Phe Arg Phe Cys Asp Thr Val Gly Val Leu Asp Pro Phe Ser Thr Tyr
165 170 175
Ala Lys Val Lys Gln Leu Val Ser Ala Leu Ser Ile Pro Leu Glu Ile
180 185 190
His Thr His Asn Asp Phe Gly Leu Ala Thr Ala Asn Ala Leu Ala Gly
195 200 205
Ile Lys Ala Gly Ala Thr Ser Val Asn Thr Thr Val Asn Gly Val Gly
210 215 220
Glu Arg Ala Gly Asn Ala Ala Leu Glu Glu Val Val Met Ser Ile Lys
225 230 235 240
Arg Ile Tyr Gly Ile Asn Leu Gly Ile Asp Thr Arg Arg Leu Leu Glu
245 250 255
Leu Ser Gln Leu Val Ala Ser Ala Ser Asn Cys His Val Pro Pro Trp
260 265 270
Lys Ala Ile Val Gly Glu Asn Thr Phe Ala His Glu Ser Gly Ile His
275 280 285
Ala His Gly Val Leu Gln Asn Pro Leu Thr Tyr Glu Pro Phe Ala Pro
290 295 300
Glu Glu Val Gly Trp Glu Arg Arg Leu Val Val Gly Lys His Ser Gly
305 310 315 320
Arg His Leu Val Thr Ser Leu Leu Gln Gln Asn Asp Ile Phe Leu Asn
325 330 335
Pro Glu Glu Thr Gln Ser Val Leu Asp Ala Val Arg Gln Gln Ser Val
340 345 350
Lys Gln Lys Arg Asn Leu Thr Val Glu Glu Leu Leu Asn Leu Val Arg
355 360 365
Glu Gln Arg Tyr Ser His Ala Thr
370 375
<210> 210
<211> 376
<212> PRT
<213> Methanosarcina acetivorans
<400> 210
Met Lys Ala Asp Ile Lys Val Ser Ile Val Asp Gln Thr Ile Asn Glu
1. 5 10 15
Val Val Arg Leu Gly Val Asn Asn Pro Ala Asp Val Arg Phe Met Leu
20 25 30
Ser Val Leu Lys Lys Tyr Ser Phe Asp Ala Ala Asp Val Ser Leu Asn
35 40 45
Asn Leu Glu Lys Asn Met Val Glu Phe Glu Ala Asp Glu Phe Ser Glu
50 55 60
Ser Met Arg Cys Arg Val Lys Cys Ser Gly Gln Glu Ile Phe Arg Ala
65 70 75 80
Lys Lys Leu Gly Phe Ser Lys Ile Val Ile Asn Thr Ser Leu Asn Pro
85 90 95
Leu Thr Pro Ile Gln Asp Met Leu Glu Pro Val Leu Gln Met Ala Cys
100 105 110
Ser Asn Asp Gln Glu Ile Tyr Leu Ser Ile Asp Asn Ala Leu Glu Phe
115 120 125
Ser Ile Arg Asp Val Glu Thr Ile Tyr Pro Leu Ile Pro Lys Tyr Gly
130 135 140
Ile Lys Arg Leu Ile Leu Gly Asp Arg Ser Gly Lys Ala Asp Pro Phe
145 150 155 160
Thr Thr Tyr Asp Lys Leu Gly Phe Leu Gly Asn Thr Ile Gln Cys Pro
165 170 175
Val Glu Tyr Val Gly Tyr Asn Asp Tyr Gly Thr Ala Thr Ala Asn Thr
180 185 190
Leu Ser Ala Leu Arg Ala Gly Ile Glu Tyr Val Ala Thr Ala Val Ser
195 200 205
Gly Ile Gly Ile Pro Gly Val Ala Ala Met Glu Glu Val Leu Met Ala
210 215 220
Ala Arg His Leu Trp Lys Asn Glu Leu Val Pro Asp Gly Tyr Ser Ile
225 230 235 240
Ala Ala Asp Cys Glu Asn Ile Leu Tyr Arg Ala Gly Ile Met Leu Pro
245 250 255
Gly Glu Lys Ala Ile Ile Gly Lys Asn Val Phe Ala His Glu Ser Gly
260 265 270
Ile His Val Asp Gly Val Leu Lys Asn Pro Asn Leu Tyr Glu Ala Ile
275 280 285
Lys Pro Glu Glu Val Gly Leu Arg Arg Leu Leu Val Ile Gly Lys His
290 295 300
Ser Gly Thr Ala Ser Leu Val Gln Lys Leu Arg His Leu Gly Leu Ser
305 310 315 320
Leu Ser Pro Glu Lys Ala Ala Ala Leu Leu Glu Lys Val Arg Asn Thr
325 330 335
Ala Ile Leu Gln Lys Lys Pro Leu Thr Asp Leu Gln Leu Lys Thr Leu
340 345 350
Tyr Asp Leu Gln Met Glu Ser Val Lys Asp Pro Asn Ile His Leu Ser
355 360 365
Gly Lys Gly Glu Met Pro Cys Asp
370 375
<210> 211
<211> 376
<212> PRT
<213> Chlorobaculum tepidum
<400> 211
Met Ile Arg Lys Pro Trp Ile Ile Asp Thr Thr Leu Arg Asp Gly Glu
1. 5 10 15
Gln Ala Pro Gly Val Val Phe Ser Pro His Glu Lys Lys Arg Ile Ala
20 25 30
Ala Met Leu Ala Glu Thr Gly Val Asp Glu Ile Glu Val Gly Tyr Pro
35 40 45
Ala Ile Ser Ala Ala Glu Arg Lys Val Ile Arg Glu Ile Val Ala Met
50 55 60
Lys Leu Pro Val Arg Leu Thr Ser Trp Ser Arg Ala Asn Met Ala Asp
65 70 75 80
Ile Glu Leu Ala Ala Glu Cys Gly Thr Asp Ala Val His Ile Ser Phe
85 90 95
Pro Ala Ser Arg Leu Tyr Leu Glu Leu Ile His Lys Lys Asp Asp Trp
100 105 110
Ile Gln Glu Gln Leu His Ala Leu Val Ser Lys Ala Arg Glu Arg Phe
115 120 125
Asp Phe Val Ser Val Gly Gly Gln Asp Ala Thr Arg Ser Ser Thr Asp
130 135 140
Phe Leu Gln Arg Phe Met Leu Asp Ala Glu Ala Ala Gly Ala Lys Arg
145 150 155 160
Phe Arg Ile Ala Asp Thr Val Gly Ile Ala Thr Pro Val Ser Val Met
165 170 175
Ala Leu Gly Ala Ala Leu Arg Gln Ser Ser Ser Leu Pro Leu Glu Phe
180 185 190
His Ala His Asn Asp Leu Gly Met Ala Thr Ala Asn Ala Phe Thr Ala
195 200 205
Leu Asn Glu Gly Phe Glu Ala Val Ser Val Ser Val Thr Gly Leu Gly
210 215 220
Glu Arg Ala Gly Asn Ala Ala Leu Glu Glu Leu Ala Met Ala Leu Ala
225 230 235 240
Leu Asn Gly Asp Phe Asp Thr His Leu Asp Thr Ser Met Leu Ser Arg
245 250 255
Leu Cys Asp Ala Val Ala Thr Ala Ser Gly Arg Ala Ile Gln Glu Gln
260 265 270
Lys Pro Val Val Gly Arg Ser Ala Phe Gln His Glu Ser Gly Ile His
275 280 285
Cys Ala Ala Leu Leu Gln Asp Pro Leu Ser Tyr Gln Pro Phe Leu Pro
290 295 300
Ser Arg Val Gly Arg Ser Asp Phe Glu Ile Val Ile Gly Lys His Ser
305 310 315 320
Gly Thr Ala Ala Ile Ile Ala His Phe Asn Arg Arg Gly Ile Thr Ile
325 330 335
Ser Lys Lys Glu Ala Arg Glu Leu Leu Asp Leu Ile Arg Ser Gln Ser
340 345 350
Asp Arg Leu Lys Arg Ala Leu Arg Thr Asp Glu Ile Asp Ala Leu Arg
355 360 365
Glu Gln Asn Ser Val Lys His Ala
370 375
<210> 212
<211> 511
<212> PRT
<213> Methanocaldococcus infernus
<400> 212
Met Ile Leu Tyr Lys Glu Glu Asn Glu Ile Ile Lys Glu Ala Leu Lys
1. 5 10 15
Gly Leu Lys Leu Pro Asp Arg Val Tyr Ile Phe Asp Thr Thr Leu Arg
20 25 30
Asp Gly Glu Gln Thr Pro Gly Val Ser Leu Thr Val Asp Glu Lys Val
35 40 45
Glu Ile Ala Ile Asn Leu Asp Lys Leu Gly Val Asp Ile Ile Glu Ala
50 55 60
Gly Phe Pro Ile Ser Ser Ser Gly Glu Tyr Glu Ala Val Lys Lys Ile
65 70 75 80
Ala Ser Leu Asn Leu Asp Ala Glu Ile Cys Ala Leu Ala Arg Ala Val
85 90 95
Lys Glu Asp Ile Asp Arg Ala Ile Asp Cys Gly Val Asp Arg Ile His
100 105 110
Thr Phe Ile Ala Thr Ser Pro Leu His Arg Lys Tyr Lys Leu Lys Met
115 120 125
Ser Lys Glu Glu Ile Val Glu Lys Ala Val Asn Ala Ile Glu Tyr Ile
130 135 140
Lys Glu His Gly Ile Lys Val Glu Phe Ser Ala Glu Asp Ala Thr Arg
145 150 155 160
Thr Glu Ile Asp Tyr Leu Lys Glu Val Tyr Lys Lys Ala Val Glu Ala
165 170 175
Gly Ala Asp Ile Ile Asn Val Pro Asp Thr Val Gly Val Met Ile Pro
180 185 190
Arg Ala Thr Tyr Tyr Leu Ile Ser Glu Leu Arg Lys Glu Ile Asp Asn
195 200 205
Ile Ser Val His Cys His Asn Asp Phe Gly Leu Ala Val Ala Asn Ser
210 215 220
Leu Ala Ala Val Glu Ala Gly Ala Ile Gln Cys His Val Thr Val Asn
225 230 235 240
Gly Leu Gly Glu Arg Gly Gly Asn Ala Ala Leu Glu Glu Val Val Thr
245 250 255
Ser Leu His Phe Ile Tyr Gly Ile Lys Thr Lys Val Lys Thr Glu Glu
260 265 270
Leu Tyr Asn Ile Ser Lys Leu Val Glu Lys Leu Thr Glu Val Lys Val
275 280 285
Gln Pro Asn Lys Ala Val Val Gly Asp Asn Ala Phe Ala His Glu Ser
290 295 300
Gly Ile His Ala His Gly Val Leu Ala His Ala Leu Thr Tyr Glu Pro
305 310 315 320
Ile Pro Pro Glu Leu Val Gly Gln Arg Arg Arg Ile Ile Leu Gly Lys
325 330 335
His Thr Gly Thr His Ala Ile Glu Ala Lys Leu Lys Glu Leu Gly Tyr
340 345 350
Thr Asn Ile Asn Lys Glu Gln Phe Lys Glu Ile Val Lys Arg Ile Lys
355 360 365
Ser Leu Gly Asp Lys Gly Lys Arg Val Thr Asp Lys Asp Val Glu Ala
370 375 380
Ile Val Glu Asp Val Ile Gly Arg Val Ser Lys Arg Glu Arg Val Val
385 390 395 400
Asp Leu Glu Gln Ile Ala Val Met Thr Gly Asn Lys Val Ile Pro Thr
405 410 415
Ala Ser Val Ala Leu Lys Ile Asn Asp Asn Leu Ile Lys Thr Ser Ala
420 425 430
Ile Gly Val Gly Pro Val Asp Ala Ala Val Lys Ala Ile Gln Lys Ala
435 440 445
Ile Gly Glu Lys Ile Lys Ile Lys Glu Tyr His Ile Asp Ala Ile Thr
450 455 460
Gly Gly Thr Asp Ala Leu Ala Glu Val Val Val Thr Leu Glu Gly Tyr
465 470 475 480
Gly Lys Glu Ile Thr Thr Lys Ala Ala Arg Glu Asp Ile Val Arg Ala
485 490 495
Ser Val Glu Ala Val Ile Asp Gly Ile Asn Lys Ile Leu Lys Lys
500 505 510
<210> 213
<211> 490
<212> PRT
<213> Methanocaldococcus infernus
<400> 213
Met Lys Val Arg Val Phe Asp Thr Thr Leu Arg Asp Gly Glu Gln Thr
1. 5 10 15
Pro Gly Val Ser Leu Thr Pro Ser Asp Lys Leu Glu Ile Ala Lys Ala
20 25 30
Leu Asp Glu Leu Gly Val Asp Val Ile Glu Ala Gly Ser Ala Ile Thr
35 40 45
Ser Lys Gly Glu Arg Glu Gly Ile Lys Leu Ile Thr Arg Glu Asn Leu
50 55 60
Asn Ala Glu Ile Cys Ser Phe Val Arg Pro Leu Thr Val Asp Ile Asp
65 70 75 80
Ala Ala Ile Glu Cys Glu Val Asp Ser Ile His Leu Val Val Pro Ser
85 90 95
Ser Pro Ile His Ile Lys Tyr Lys Leu Lys Lys Ser Glu Asp Glu Val
100 105 110
Leu Asp Leu Ala Val Asn Ala Ile Glu Tyr Ala Lys Asp His Gly Leu
115 120 125
Ile Val Glu Leu Ser Ala Glu Asp Ala Thr Arg Ala Glu Leu Asn Phe
130 135 140
Leu Ile Lys Leu Phe Lys Ala Gly Glu Asn Leu Ala Asp Arg Val Cys
145 150 155 160
Val Cys Asp Thr Val Gly Val Leu Thr Pro Gln Lys Ser Glu Glu Leu
165 170 175
Phe Lys Lys Ile Thr Ser Glu Ile Lys Leu Pro Val Ser Val His Cys
180 185 190
His Asn Asp Phe Gly Met Ala Thr Ala Asn Thr Cys Ser Ala Ile Leu
195 200 205
Gly Gly Ala Val Gln Cys His Val Thr Val Asn Gly Ile Gly Glu Arg
210 215 220
Ala Gly Asn Ala Ala Leu Glu Glu Val Val Thr Ala Leu Lys Phe Leu
225 230 235 240
Tyr Asn Ile Glu Thr Asn Ile Lys Leu Glu Lys Leu Tyr Glu Val Ser
245 250 255
Arg Leu Val Ala Arg Leu Met Lys Leu Pro Val Pro Pro Asn Lys Ala
260 265 270
Ile Val Gly Asp Asn Ala Phe Ala His Glu Ala Gly Ile His Val Asp
275 280 285
Gly Leu Ile Lys Asn Thr Lys Thr Tyr Glu Pro Ile Ser Pro Glu Val
290 295 300
Val Gly Asn Lys Arg Arg Ile Ile Leu Gly Lys His Ser Gly Arg Lys
305 310 315 320
Ala Leu Ile Tyr Lys Leu Lys Leu Met Gly Ile Glu Ala Ser Glu Glu
325 330 335
Gln Ile Asn Lys Ile Tyr Glu Lys Ile Lys Glu Leu Gly Asp Leu Gly
340 345 350
Lys Tyr Val Ser Asp Ala Asp Leu Met Ala Ile Val Lys Asp Val Leu
355 360 365
Gly Lys Asp Leu Glu Glu Lys Ile Glu Leu Asp Glu Leu Thr Val Val
370 375 380
Ser Gly Asn Lys Ile Thr Pro Ile Ala Ser Val Lys Leu His Tyr Lys
385 390 395 400
Gly Glu Asp Arg Leu Leu Ile Glu Thr Ala Tyr Gly Val Gly Pro Val
405 410 415
Asp Ala Ala Ile Asn Ala Val Arg Lys Ala Ile Ser Gly Val Ala Asp
420 425 430
Ile Lys Leu Glu Glu Tyr Lys Val Glu Ala Ile Gly Gly Gly Thr Asp
435 440 445
Ala Ile Ile Glu Val Thr Val Lys Leu Arg Lys Gly Val Asn Thr Val
450 455 460
Glu Val Lys Lys Ala Asp Ser Asp Ile Ile Arg Ala Ser Val Asn Ala
465 470 475 480
Val Met Glu Gly Ile Asn Leu Leu Leu Gln
485 490
<210> 214
<211> 396
<212> PRT
<213> Methanocaldococcus infernus
<400> 214
Met Asp Leu Leu Tyr Glu Asn Thr Trp Lys Ala Pro Ser Pro Tyr Asn
1. 5 10 15
Pro Lys Leu Lys Leu Lys Asp Ile Tyr Ile Tyr Asp Thr Thr Leu Arg
20 25 30
Asp Gly Glu Gln Thr Pro Gly Val Cys Phe Thr Lys Glu Gln Lys Leu
35 40 45
Glu Ile Ala Arg Ala Leu Asp Glu Leu Gly Val Ser Gln Ile Glu Ala
50 55 60
Gly Phe Pro Ile Val Ser Lys Arg Glu Ala Glu Ile Val Lys Ala Ile
65 70 75 80
Ala Ser Glu Asn Leu Asn Ala Asp Ile Leu Ala Leu Ser Arg Ile Arg
85 90 95
Lys Glu Asp Ile Asn Lys Ala Ile Asp Cys Asp Val Asp Gly Ile Ile
100 105 110
Thr Phe Ile Ala Thr Ser Pro Leu His Ile Lys Cys Lys Phe Lys Gly
115 120 125
Lys Arg Leu Glu Asp Phe Phe Asp Thr Ile Val Glu Cys Ile Glu Tyr
130 135 140
Ala Lys Ser His Gly Leu Phe Val Ala Phe Ser Ala Glu Asp Gly Thr
145 150 155 160
Arg Thr Pro Leu Glu Asp Leu Ile Arg Val His Lys Leu Ala Glu Glu
165 170 175
Ala Gly Ala Asp Arg Val His Val Ala Asp Thr Ala Gly Thr Ala Thr
180 185 190
Pro Gln Ala Met Glu Phe Ile Cys Lys Ala Leu Thr Cys Ser Leu Asn
195 200 205
Lys Ala His Val Gly Val His Cys His Asn Asp Phe Gly Leu Ala Val
210 215 220
Ile Asn Ser Ile Tyr Gly Leu Ile Gly Gly Ala Lys Ala Val Ser Thr
225 230 235 240
Thr Val Asn Gly Ile Gly Glu Arg Ala Gly Asn Thr Ser Leu Glu Glu
245 250 255
Leu Ile Met Ser Leu Ile Val Leu Tyr Asp Val Asp Leu Lys Leu Asn
260 265 270
Leu Glu Val Leu Pro Lys Leu Cys Arg Met Val Glu Glu Tyr Ser Gly
275 280 285
Ile Lys Asn Pro Lys Asn Lys Pro Ile Val Gly Glu Leu Val Phe Ser
290 295 300
His Glu Ser Gly Ile His Val Asp Ala Val Ile Glu Asn Pro Leu Thr
305 310 315 320
Tyr Glu Pro Phe Leu Pro Glu Lys Ile Gly Leu Lys Arg Asn Ile Ile
325 330 335
Leu Gly Lys His Ser Gly Lys Arg Ala Val Lys Tyr Lys Leu Lys Leu
340 345 350
Leu Gly Val Glu Val Glu Asp Lys Leu Leu Asp Lys Ile Val Glu Arg
355 360 365
Val Lys Glu Leu Arg Glu Lys Gly Glu Lys Ile Asp Asp Glu Lys Leu
370 375 380
Leu Glu Ile Val Glu Glu Ile Lys Arg Ile Lys Asp
385 390 395
<210> 215
<211> 540
<212> PRT
<213> Lotus japonicus
<400> 215
Met Ala Ser Lys Asn Ser Ile Ile Arg Asn Arg Pro Glu Tyr Ile Pro
1. 5 10 15
Asn His Ile Pro Asn Pro Thr Tyr Val Arg Ile Leu Asp Thr Thr Leu
20 25 30
Arg Asp Gly Glu Gln Ser Pro Gly Ala Ala Met Thr Cys Val Gln Lys
35 40 45
Leu Glu Thr Ala Arg Gln Leu Ala Lys Leu Gly Val Asp Ile Ile Glu
50 55 60
Ala Gly Phe Pro Cys Ala Ser Lys Gln Asp Phe Met Ala Val Lys Met
65 70 75 80
Ile Ala Glu Glu Val Gly Asn Cys Val Asp Gly Asn Gly Tyr Val Pro
85 90 95
Val Ile Thr Gly Val Ser Arg Cys Asn Glu Lys Asp Ile Ala Thr Ala
100 105 110
Trp Glu Ala Leu Lys His Ala Lys Arg Pro Arg Leu Arg Thr Phe Ile
115 120 125
Ala Thr Ser Pro Ile His Met Glu Tyr Lys Leu Arg Lys Ser Lys Asp
130 135 140
Gln Val Leu Glu Thr Ala Arg Asn Met Val Lys Phe Ala Arg Ser Leu
145 150 155 160
Gly Cys Thr Asp Ile Gln Phe Gly Ala Glu Asp Ala Ala Arg Ser Asp
165 170 175
Lys Glu Phe Leu Tyr Gln Ile Phe Gly Glu Val Ile Lys Ala Gly Ala
180 185 190
Thr Thr Leu Thr Ile Pro Asp Thr Val Gly Ile Ala Met Pro Phe Glu
195 200 205
Tyr Gly Lys Leu Ile Ala Asp Ile Lys Ala Asn Thr Pro Gly Ile Glu
210 215 220
Asn Ala Ile Met Ala Thr His Cys His Asn Asp Leu Gly Leu Ala Thr
225 230 235 240
Ala Asn Thr Ile Glu Gly Ala Arg Tyr Gly Ala Arg Gln Leu Glu Val
245 250 255
Thr Ile Asn Gly Ile Gly Glu Arg Ala Gly Asn Ala Ser Phe Glu Glu
260 265 270
Val Val Met Ala Leu Thr Cys Arg Gly Ile Asp Ile Leu Gly Gly Leu
275 280 285
His Thr Gly Ile Asn Thr Arg His Ile Leu Lys Thr Ser Lys Met Val
290 295 300
Glu Lys Tyr Ser Gly Leu His Leu Gln Pro His Lys Ala Leu Val Gly
305 310 315 320
Ala Asn Ala Phe Leu His Glu Ser Gly Ile His Gln Asp Gly Met Leu
325 330 335
Lys His Arg Gly Thr Tyr Glu Ile Ile Ser Pro Glu Asp Ile Gly Leu
340 345 350
Val Arg Ser Val Gly Asp Thr Ile Val Leu Gly Lys Leu Ser Gly Arg
355 360 365
Gln Ala Leu Arg Asn Arg Leu Glu Glu Leu Gly Tyr Lys Leu Lys Asp
370 375 380
Thr Glu Val Glu Gly Val Phe Trp Gln Phe Lys Ala Val Ala Glu Lys
385 390 395 400
Lys Lys Arg Ile Thr Asp Thr Asp Leu Arg Ala Leu Val Ser Asn Glu
405 410 415
Ala Phe Asn Glu Gln Pro Ile Trp Lys Leu Gly Asp Leu Gln Val Thr
420 425 430
Cys Gly Thr Val Gly Phe Ser Thr Ala Thr Val Lys Leu Phe Ser Ile
435 440 445
Asp Gly Ser Met His Val Ala Cys Ser Ile Gly Thr Gly Pro Val Asp
450 455 460
Ser Ala Tyr Lys Ala Ile Asn His Ile Val Lys Glu Pro Ala Lys Leu
465 470 475 480
Val Lys Tyr Thr Leu Gly Ala Ile Thr Glu Gly Ile Asp Ala Thr Ala
485 490 495
Thr Thr Ser Val Glu Ile Ser Arg Gly Asp Thr Asn His Pro Val Phe
500 505 510
Ser Gly Thr Gly Gly Gly Thr Asp Val Val Val Ser Ser Val Asp Ala
515 520 525
Tyr Leu Ser Ala Leu Asn Asn Met Leu Arg Phe Tyr
530 535 540
<210> 216
<211> 518
<212> PRT
<213> Azotobacter vinelandii
<400> 216
Met Pro His His Glu Phe Glu Cys Ser Lys Val Ile Pro Glu Arg Lys
1. 5 10 15
Lys His Ala Val Ile Lys Gly Lys Gly Glu Thr Leu Ala Asp Ala Leu
20 25 30
Pro Gln Gly Tyr Leu Asn Thr Ile Pro Gly Ser Ile Ser Glu Arg Gly
35 40 45
Cys Ala Tyr Cys Gly Ala Lys His Val Ile Gly Thr Pro Met Lys Asp
50 55 60
Val Ile His Ile Ser His Gly Pro Val Gly Cys Thr Tyr Asp Thr Trp
65 70 75 80
Gln Thr Lys Arg Tyr Ile Ser Asp Asn Asp Asn Phe Gln Leu Lys Tyr
85 90 95
Thr Tyr Ala Thr Asp Val Lys Glu Lys His Ile Val Phe Gly Ala Glu
100 105 110
Lys Leu Leu Lys Gln Asn Ile Ile Glu Ala Phe Lys Ala Phe Pro Gln
115 120 125
Ile Lys Arg Met Thr Ile Tyr Gln Thr Cys Ala Thr Ala Leu Ile Gly
130 135 140
Asp Asp Ile Asn Ala Ile Ala Glu Glu Val Met Glu Glu Met Pro Glu
145 150 155 160
Val Asp Ile Phe Val Cys Asn Ser Pro Gly Phe Ala Gly Pro Ser Gln
165 170 175
Ser Gly Gly His His Lys Ile Asn Ile Ala Trp Ile Asn Gln Lys Val
180 185 190
Gly Thr Val Glu Pro Glu Ile Thr Gly Asp His Val Ile Asn Tyr Val
195 200 205
Gly Glu Tyr Asn Ile Gln Gly Asp Gln Glu Val Met Val Asp Tyr Phe
210 215 220
Lys Arg Met Gly Ile Gln Val Leu Ser Thr Phe Thr Gly Asn Gly Ser
225 230 235 240
Tyr Asp Gly Leu Arg Ala Met His Arg Ala His Leu Asn Val Leu Glu
245 250 255
Cys Ala Arg Ser Ala Glu Tyr Ile Cys Asn Glu Leu Arg Val Arg Tyr
260 265 270
Gly Ile Pro Arg Leu Asp Ile Asp Gly Phe Gly Phe Lys Pro Leu Ala
275 280 285
Asp Ser Leu Arg Lys Ile Gly Met Phe Phe Gly Ile Glu Asp Arg Ala
290 295 300
Lys Ala Ile Ile Asp Glu Glu Val Ala Arg Trp Lys Pro Glu Leu Asp
305 310 315 320
Trp Tyr Lys Glu Arg Leu Met Gly Lys Lys Val Cys Leu Trp Pro Gly
325 330 335
Gly Ser Lys Leu Trp His Trp Ala His Val Ile Glu Glu Glu Met Gly
340 345 350
Leu Lys Val Val Ser Val Tyr Thr Lys Phe Gly His Gln Gly Asp Met
355 360 365
Glu Lys Gly Ile Ala Arg Cys Gly Glu Gly Thr Leu Ala Ile Asp Asp
370 375 380
Pro Asn Glu Leu Glu Gly Leu Glu Ala Leu Glu Met Leu Lys Pro Asp
385 390 395 400
Ile Ile Leu Thr Gly Lys Arg Pro Gly Glu Val Ala Lys Lys Val Arg
405 410 415
Val Pro Tyr Leu Asn Ala His Ala Tyr His Asn Gly Pro Tyr Lys Gly
420 425 430
Phe Glu Gly Trp Val Arg Phe Ala Arg Asp Ile Tyr Asn Ala Ile Tyr
435 440 445
Ser Pro Ile His Gln Leu Ser Gly Ile Asp Ile Thr Lys Asp Asn Ala
450 455 460
Pro Glu Trp Gly Asn Gly Phe Arg Thr Arg Gln Met Leu Ser Asp Gly
465 470 475 480
Asn Leu Ser Asp Ala Val Arg Asn Ser Glu Thr Leu Arg Gln Tyr Thr
485 490 495
Gly Gly Tyr Asp Ser Val Ser Lys Leu Arg Glu Arg Glu Tyr Pro Ala
500 505 510
Phe Glu Arg Lys Val Gly
515
<210> 217
<211> 462
<212> PRT
<213> Azotobacter vinelandii
<400> 217
Met Thr Cys Glu Val Lys Glu Lys Gly Arg Val Gly Thr Ile Asn Pro
1. 5 10 15
Ile Phe Thr Cys Gln Pro Ala Gly Ala Gln Phe Val Ser Ile Gly Ile
20 25 30
Lys Asp Cys Ile Gly Ile Val His Gly Gly Gln Gly Cys Val Met Phe
35 40 45
Val Arg Leu Ile Phe Ser Gln His Tyr Lys Glu Ser Phe Glu Leu Ala
50 55 60
Ser Ser Ser Leu His Glu Asp Gly Ala Val Phe Gly Ala Cys Gly Arg
65 70 75 80
Val Glu Glu Ala Val Asp Val Leu Leu Ser Arg Tyr Pro Asp Val Lys
85 90 95
Val Val Pro Ile Ile Thr Thr Cys Ser Thr Glu Ile Ile Gly Asp Asp
100 105 110
Val Asp Gly Val Ile Lys Lys Leu Asn Glu Gly Leu Leu Lys Glu Lys
115 120 125
Phe Pro Asp Arg Glu Val His Leu Ile Ala Met His Thr Pro Ser Phe
130 135 140
Val Gly Ser Met Ile Ser Gly Tyr Asp Val Ala Val Arg Asp Val Val
145 150 155 160
Arg His Phe Ala Lys Arg Glu Ala Pro Asn Asp Lys Ile Asn Leu Leu
165 170 175
Thr Gly Trp Val Asn Pro Gly Asp Val Lys Glu Leu Lys His Leu Leu
180 185 190
Gly Glu Met Asp Ile Glu Ala Asn Val Leu Phe Glu Ile Glu Ser Phe
195 200 205
Asp Ser Pro Ile Leu Pro Asp Gly Ser Ala Val Ser His Gly Asn Thr
210 215 220
Thr Ile Glu Asp Leu Ile Asp Thr Gly Asn Ala Arg Ala Thr Phe Ala
225 230 235 240
Leu Asn Arg Tyr Glu Gly Thr Lys Ala Ala Glu Tyr Leu Gln Lys Lys
245 250 255
Phe Glu Ile Pro Ala Ile Ile Gly Pro Thr Pro Ile Gly Ile Arg Asn
260 265 270
Thr Asp Ile Phe Leu Gln Asn Leu Lys Lys Ala Thr Gly Lys Pro Ile
275 280 285
Pro Gln Ser Leu Ala His Glu Arg Gly Val Ala Ile Asp Ala Leu Ala
290 295 300
Asp Leu Thr His Met Phe Leu Ala Glu Lys Arg Val Ala Ile Tyr Gly
305 310 315 320
Ala Pro Asp Leu Val Ile Gly Leu Ala Glu Phe Cys Leu Asp Leu Glu
325 330 335
Met Lys Pro Val Leu Leu Leu Leu Gly Asp Asp Asn Ser Lys Tyr Val
340 345 350
Asp Asp Pro Arg Ile Lys Ala Leu Gln Glu Asn Val Asp Tyr Gly Met
355 360 365
Glu Ile Val Thr Asn Ala Asp Phe Trp Glu Leu Glu Asn Arg Ile Lys
370 375 380
Asn Glu Gly Leu Glu Leu Asp Leu Ile Leu Gly His Ser Lys Gly Arg
385 390 395 400
Phe Ile Ser Ile Asp Tyr Asn Ile Pro Met Leu Arg Val Gly Phe Pro
405 410 415
Thr Tyr Asp Arg Ala Gly Leu Phe Arg Tyr Pro Thr Val Gly Tyr Gly
420 425 430
Gly Ala Ile Trp Leu Ala Glu Gln Met Ala Asn Thr Leu Phe Ala Asp
435 440 445
Met Glu His Lys Lys Asn Lys Glu Trp Val Leu Asn Val Trp
450 455 460
<210> 218
<211> 275
<212> PRT
<213> Azotobacter vinelandii
<400> 218
Met Thr Arg Lys Val Ala Ile Tyr Gly Lys Gly Gly Ile Gly Lys Ser
1. 5 10 15
Thr Thr Thr Gln Asn Thr Ala Ala Ala Leu Ala Tyr Phe His Asp Lys
20 25 30
Lys Val Phe Ile His Gly Cys Asp Pro Lys Ala Asp Ser Thr Arg Leu
35 40 45
Ile Leu Gly Gly Lys Pro Gln Glu Thr Leu Met Asp Met Leu Arg Asp
50 55 60
Lys Gly Ala Glu Lys Ile Thr Asn Asp Asp Val Ile Lys Lys Gly Phe
65 70 75 80
Leu Asp Ile Gln Cys Val Glu Ser Gly Gly Pro Glu Pro Gly Val Gly
85 90 95
Cys Ala Gly Arg Gly Val Ile Thr Ala Ile Asp Leu Met Glu Glu Asn
100 105 110
Gly Ala Tyr Thr Asp Asp Leu Asp Phe Val Phe Phe Asp Val Leu Gly
115 120 125
Asp Val Val Cys Gly Gly Phe Ala Met Pro Ile Arg Asp Gly Lys Ala
130 135 140
Gln Glu Val Tyr Ile Val Ala Ser Gly Glu Met Met Ala Ile Tyr Ala
145 150 155 160
Ala Asn Asn Ile Cys Lys Gly Leu Val Lys Tyr Ala Lys Gln Ser Gly
165 170 175
Val Arg Leu Gly Gly Ile Ile Cys Asn Ser Arg Lys Val Asp Gly Glu
180 185 190
Arg Glu Phe Leu Glu Glu Phe Thr Ala Ala Ile Gly Thr Lys Met Ile
195 200 205
His Phe Val Pro Arg Asp Asn Ile Val Gln Lys Ala Glu Phe Asn Lys
210 215 220
Lys Thr Val Thr Glu Phe Ala Pro Glu Glu Asn Gln Ala Lys Glu Tyr
225 230 235 240
Gly Glu Leu Ala Arg Lys Ile Ile Glu Asn Asp Glu Phe Val Ile Pro
245 250 255
Lys Pro Leu Thr Met Asp Gln Leu Glu Asp Met Val Val Lys Tyr Gly
260 265 270
Ile Ala Asp
275
<210> 219
<211> 132
<212> PRT
<213> Azotobacter vinelandii
<400> 219
Met Ser Thr Ala Ser Ala Ala Ala Val Val Lys Gln Lys Val Glu Ala
1. 5 10 15
Pro Val His Pro Met Asp Ala Arg Ile Asp Glu Leu Thr Asp Tyr Ile
20 25 30
Met Lys Asn Cys Leu Trp Gln Phe His Ser Arg Ser Trp Asp Arg Glu
35 40 45
Arg Gln Asn Ala Glu Ile Leu Lys Lys Thr Lys Glu Leu Leu Cys Gly
50 55 60
Glu Pro Val Asp Leu Ser Thr Ser His Asp Arg Cys Tyr Trp Val Asp
65 70 75 80
Ala Val Cys Leu Ala Asp Asp Tyr Arg Glu His Tyr Pro Trp Ile Asn
85 90 95
Ser Met Ser Lys Glu Glu Ile Gly Ser Leu Met Gln Gly Leu Lys Asp
100 105 110
Arg Met Asp Tyr Leu Thr Ile Thr Gly Ser Leu Asn Glu Glu Leu Ser
115 120 125
Asp Lys His Tyr
130
<210> 220
<211> 4
<212> PRT
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 220
Arg Arg Asn Phe
1
<210> 221
<211> 606
<212> PRT
<213> Nicotiana benthamiana
<400> 221
Met Ala Ser Ile Thr Thr Asn His Thr Phe Ser Arg Asn Leu Asn Phe
1. 5 10 15
Ser Phe His Pro Gln Asn Pro Leu Ile Gln Thr Gln Ala Leu Phe Lys
20 25 30
Phe Lys Pro Ser Ile Pro Asn Cys Ser Pro Ile Ile Arg Cys Ala Ile
35 40 45
Arg Arg Arg Pro Glu Tyr Thr Pro Ser His Ile Pro Asp Pro Lys Tyr
50 55 60
Ile Arg Ile Phe Asp Thr Thr Leu Arg Asp Gly Glu Gln Ser Pro Gly
65 70 75 80
Ala Thr Met Thr Thr Lys Glu Lys Leu Asp Val Ala Arg Gln Leu Ala
85 90 95
Lys Leu Gly Val Asp Ile Ile Glu Ala Gly Phe Pro Ala Ser Ser Glu
100 105 110
Ala Asp Leu Glu Ala Val Lys Leu Ile Ala Lys Glu Val Gly Asn Gly
115 120 125
Val Tyr Glu Glu Gly His Val Pro Val Ile Cys Gly Leu Ala Arg Cys
130 135 140
Asn Lys Arg Asp Ile Asp Lys Ala Trp Glu Ala Val Lys Tyr Ala Lys
145 150 155 160
Lys Pro Arg Ile His Thr Phe Ile Ala Thr Ser Glu Ile His Met Lys
165 170 175
Phe Lys Leu Lys Met Ser Arg Asp Glu Val Val Glu Lys Ala Arg Ser
180 185 190
Met Val Ala Tyr Ala Arg Ser Ile Gly Cys Glu Asp Val Glu Phe Ser
195 200 205
Pro Glu Asp Ala Gly Arg Ser Asp Pro Glu Phe Leu Tyr His Ile Leu
210 215 220
Gly Glu Val Ile Lys Ala Gly Ala Thr Thr Leu Asn Ile Pro Asp Thr
225 230 235 240
Val Gly Tyr Thr Val Pro Ser Glu Phe Gly Lys Leu Ile Ala Asp Ile
245 250 255
Lys Ala Asn Thr Pro Gly Ile Gly Asp Val Ile Ile Ser Thr His Cys
260 265 270
Gln Asn Asp Leu Gly Leu Ser Thr Ala Asn Thr Leu Ala Gly Ala Cys
275 280 285
Ala Gly Ala Arg Gln Val Glu Val Thr Ile Asn Gly Ile Gly Glu Arg
290 295 300
Ala Gly Asn Ala Ser Leu Glu Glu Val Val Met Ala Leu Lys Cys Arg
305 310 315 320
Gly Glu Gln Val Leu Gly Gly Leu Tyr Thr Gly Ile Asn Thr Gln His
325 330 335
Ile Leu Met Ser Ser Lys Met Val Glu Glu Tyr Thr Gly Leu His Val
340 345 350
Gln Pro His Lys Ala Ile Val Gly Ala Asn Ala Phe Ala His Glu Ser
355 360 365
Gly Ile His Gln Asp Gly Met Leu Lys His Lys Asp Thr Tyr Glu Ile
370 375 380
Ile Ser Pro Glu Asp Ile Gly Leu Asn Arg Ala Asn Glu Ala Gly Ile
385 390 395 400
Val Leu Gly Lys Leu Ser Gly Arg His Ala Leu Lys Ser Lys Met Leu
405 410 415
Glu Leu Gly Tyr Asp Ile Glu Gly Lys Glu Leu Glu Asp Leu Phe Trp
420 425 430
Arg Phe Lys Ser Val Ala Glu Lys Lys Lys Lys Val Thr Asp Asp Asp
435 440 445
Ile Ile Ala Leu Met Ser Asp Glu Val Phe Gln Pro Gln Val Val Trp
450 455 460
Gln Leu Ala Asp Val Gln Ile Thr Cys Gly Ser Leu Gly Leu Ser Thr
465 470 475 480
Ala Thr Val Lys Leu Ile Asp Ser Asp Gly Gln Asp His Val Ala Cys
485 490 495
Ser Val Gly Thr Gly Pro Val Asp Ala Ala Tyr Lys Ala Val Asp Leu
500 505 510
Ile Val Lys Val Pro Ile Thr Leu Leu Glu Tyr Ser Met Asn Ala Val
515 520 525
Thr Glu Gly Ile Asp Ser Ile Ala Ser Thr Arg Val Val Ile Arg Glu
530 535 540
Glu Asp Asp His Ala Ile Thr Asn Gly Ser Ile Gly Leu Thr Leu His
545 550 555 560
Arg Thr Phe Ser Gly Thr Gly Ala Asp Met Asp Val Val Ile Ser Ser
565 570 575
Val Arg Ala Tyr Ile Gly Ala Leu Asn Lys Met Leu Ser Phe Gly Lys
580 585 590
Leu Val Ser Arg Cys Asn Asn Pro Glu Gly Ser Val Val Val
595 600 605
<210> 222
<211> 470
<212> PRT
<213> Nicotiana benthamiana
<400> 222
Met Ala Ser Ile Tyr Ala Asn Pro Thr Thr Ser Leu Asn Thr Ser Leu
1. 5 10 15
Ser Ser Tyr Ser Lys Asn Ile Phe Leu Arg Ser Val Phe Lys Phe Met
20 25 30
Pro Ser Ile Thr Lys Arg Cys His Cys Pro Tyr Thr Ser Thr Ala Val
35 40 45
Arg Cys Thr Asn Val Arg Arg Pro Arg Pro Asn Tyr Arg Pro Gly Arg
50 55 60
Phe Ser His Pro Asn Tyr Val Gly Ile Phe Asp Thr Thr Leu Arg Asp
65 70 75 80
Gly Glu Gln Ala Pro Gly Ala Ala Met Thr Ile Thr Glu Lys Leu Asp
85 90 95
Ile Ala Arg Gln Leu Ala Lys Leu Gly Val Asp Val Ile Glu Ala Gly
100 105 110
Phe Pro Ala Ala Ser Asp Ala Asp Phe Glu Leu Val Lys Leu Val Ala
115 120 125
Lys Glu Val Gly Asn Asn Val Asp Glu Glu Gly Tyr Val Pro Val Ile
130 135 140
Cys Ala Ile Gly Arg Thr Thr Lys Lys Asp Ile Asp Arg Thr Trp Glu
145 150 155 160
Ala Leu Lys Tyr Ala Lys Lys Pro Met Ile Ser Leu Phe Ile Ala Thr
165 170 175
Ser Asp Ile His Met Lys Tyr Lys Leu Lys Met Ser Lys Glu Val Val
180 185 190
Val Glu Lys Ala Arg Ser Met Val Ala Tyr Ala Lys Thr Leu Cys Glu
195 200 205
Asp Val Arg Phe Cys Val Glu Asp Gly Ala Arg Ser Asp Arg Lys Phe
210 215 220
Leu Tyr Tyr Ile Leu Gly Glu Gly Ile Lys Val Gly Ala Thr Ala Ile
225 230 235 240
Cys Val Ala Asp Thr Val Gly Ser Ser Leu Pro Thr Glu Phe Gly Arg
245 250 255
Leu Ile Ala Asp Ile Lys Ala Asn Thr Pro Gly Ile Glu Asp Val Ile
260 265 270
Ile Ser Val His Cys His Asn Asp Leu Gly Leu Ala Thr Ala Asn Thr
275 280 285
Leu Ala Gly Ala Cys Ala Gly Ala Arg Leu Val Asp Val Thr Val Asn
290 295 300
Gly Ile Gly Glu Arg Ala Gly Asn Gly Ser Leu Glu Glu Ile Val Met
305 310 315 320
Ala Leu Lys Tyr Arg Gly Glu Glu Val Leu Gly Gly Leu Tyr Ser Gly
325 330 335
Ile Asn Thr Lys His Ile Ile Ala Thr Ser Lys Met Val Glu Glu Tyr
340 345 350
Cys Gly Leu Lys Leu Gln Pro His Lys Pro Ile Val Gly Ala Asn Ala
355 360 365
Phe Ser His Glu Ser Gly Ile His Gln Asp Gly Val Leu Lys Lys Arg
370 375 380
Glu Thr Tyr Glu Phe Val Ser Pro Glu Asp Val Gly Phe Gln Arg Val
385 390 395 400
Thr Gly His Gly Ile Ile Leu Gly Lys Leu Ser Gly Arg His Ala Leu
405 410 415
Lys Ser Lys Met Phe Glu Leu Gly Tyr Glu Phe Glu Gly Lys Glu Leu
420 425 430
Asp Asp Ile Phe Arg Arg Phe Lys Ser Val Ala Glu Lys Lys Lys Lys
435 440 445
Ile Thr Glu Glu Asp Leu Arg Ala Leu Val Ser Asp Lys Val Cys Ser
450 455 460
Leu Lys Leu Leu Asp Ala
465 470
<210> 223
<211> 644
<212> PRT
<213> Mycobacterium tuberculosis
<400> 223
Met Ala Thr Ser Glu Ser Pro Asp Ala Tyr Thr Glu Ser Phe Gly Ala
1. 5 10 15
His Thr Ile Val Lys Pro Ala Gly Pro Pro Arg Val Gly Gln Pro Ser
20 25 30
Trp Asn Pro Gln Arg Ala Ser Ser Met Pro Val Asn Arg Tyr Arg Pro
35 40 45
Phe Ala Glu Glu Val Glu Pro Ile Arg Leu Arg Asn Arg Thr Trp Pro
50 55 60
Asp Arg Val Ile Asp Arg Ala Pro Leu Trp Cys Ala Val Asp Leu Arg
65 70 75 80
Asp Gly Asn Gln Ala Leu Ile Asp Pro Met Ser Pro Ala Arg Lys Arg
85 90 95
Arg Met Phe Asp Leu Leu Val Arg Met Gly Tyr Lys Glu Ile Glu Val
100 105 110
Gly Phe Pro Ser Ala Ser Gln Thr Asp Phe Asp Phe Val Arg Glu Ile
115 120 125
Ile Glu Gln Gly Ala Ile Pro Asp Asp Val Thr Ile Gln Val Leu Thr
130 135 140
Gln Cys Arg Pro Glu Leu Ile Glu Arg Thr Phe Gln Ala Cys Ser Gly
145 150 155 160
Ala Pro Arg Ala Ile Val His Phe Tyr Asn Ser Thr Ser Ile Leu Gln
165 170 175
Arg Arg Val Val Phe Arg Ala Asn Arg Ala Glu Val Gln Ala Ile Ala
180 185 190
Thr Asp Gly Ala Arg Lys Cys Val Glu Gln Ala Ala Lys Tyr Pro Gly
195 200 205
Thr Gln Trp Arg Phe Glu Tyr Ser Pro Glu Ser Tyr Thr Gly Thr Glu
210 215 220
Leu Glu Tyr Ala Lys Gln Val Cys Asp Ala Val Gly Glu Val Ile Ala
225 230 235 240
Pro Thr Pro Glu Arg Pro Ile Ile Phe Asn Leu Pro Ala Thr Val Glu
245 250 255
Met Thr Thr Pro Asn Val Tyr Ala Asp Ser Ile Glu Trp Met Ser Arg
260 265 270
Asn Leu Ala Asn Arg Glu Ser Val Ile Leu Ser Leu His Pro His Asn
275 280 285
Asp Arg Gly Thr Ala Val Ala Ala Ala Glu Leu Gly Phe Ala Ala Gly
290 295 300
Ala Asp Arg Ile Glu Gly Cys Leu Phe Gly Asn Gly Glu Arg Thr Gly
305 310 315 320
Asn Val Cys Leu Val Thr Leu Gly Leu Asn Leu Phe Ser Arg Gly Val
325 330 335
Asp Pro Gln Ile Asp Phe Ser Asn Ile Asp Glu Ile Arg Arg Thr Val
340 345 350
Glu Tyr Cys Asn Gln Leu Pro Val His Glu Arg His Pro Tyr Gly Gly
355 360 365
Asp Leu Val Tyr Thr Ala Phe Ser Gly Ser His Gln Asp Ala Ile Asn
370 375 380
Lys Gly Leu Asp Ala Met Lys Leu Asp Ala Asp Ala Ala Asp Cys Asp
385 390 395 400
Val Asp Asp Met Leu Trp Gln Val Pro Tyr Leu Pro Ile Asp Pro Arg
405 410 415
Asp Val Gly Arg Thr Tyr Glu Ala Val Ile Arg Val Asn Ser Gln Ser
420 425 430
Gly Lys Gly Gly Val Ala Tyr Ile Met Lys Thr Asp His Gly Leu Ser
435 440 445
Leu Pro Arg Arg Leu Gln Ile Glu Phe Ser Gln Val Ile Gln Lys Ile
450 455 460
Ala Glu Gly Thr Ala Gly Glu Gly Gly Glu Val Ser Pro Lys Glu Met
465 470 475 480
Trp Asp Ala Phe Ala Glu Glu Tyr Leu Ala Pro Val Arg Pro Leu Glu
485 490 495
Arg Ile Arg Gln His Val Asp Ala Ala Asp Asp Asp Gly Gly Thr Thr
500 505 510
Ser Ile Thr Ala Thr Val Lys Ile Asn Gly Val Glu Thr Glu Ile Ser
515 520 525
Gly Ser Gly Asn Gly Pro Leu Ala Ala Phe Val His Ala Leu Ala Asp
530 535 540
Val Gly Phe Asp Val Ala Val Leu Asp Tyr Tyr Glu His Ala Met Ser
545 550 555 560
Ala Gly Asp Asp Ala Gln Ala Ala Ala Tyr Val Glu Ala Ser Val Thr
565 570 575
Ile Ala Ser Pro Ala Gln Pro Gly Glu Ala Gly Arg His Ala Ser Asp
580 585 590
Pro Val Thr Ile Ala Ser Pro Ala Gln Pro Gly Glu Ala Gly Arg His
595 600 605
Ala Ser Asp Pro Val Thr Ser Lys Thr Val Trp Gly Val Gly Ile Ala
610 615 620
Pro Ser Ile Thr Thr Ala Ser Leu Arg Ala Val Val Ser Ala Val Asn
625 630 635 640
Arg Ala Ala Arg
<210> 224
<211> 290
<212> PRT
<213> Azotobacter vinelandii
<400> 224
Met Ala Met Arg Gln Cys Ala Ile Tyr Gly Lys Gly Gly Ile Gly Lys
1. 5 10 15
Ser Thr Thr Thr Gln Asn Leu Val Ala Ala Leu Ala Glu Met Gly Lys
20 25 30
Lys Val Met Ile Val Gly Cys Asp Pro Lys Ala Asp Ser Thr Arg Leu
35 40 45
Ile Leu His Ser Lys Ala Gln Asn Thr Ile Met Glu Met Ala Ala Glu
50 55 60
Ala Gly Thr Val Glu Asp Leu Glu Leu Glu Asp Val Leu Lys Ala Gly
65 70 75 80
Tyr Gly Gly Val Lys Cys Val Glu Ser Gly Gly Pro Glu Pro Gly Val
85 90 95
Gly Cys Ala Gly Arg Gly Val Ile Thr Ala Ile Asn Phe Leu Glu Glu
100 105 110
Glu Gly Ala Tyr Glu Asp Asp Leu Asp Phe Val Phe Tyr Asp Val Leu
115 120 125
Gly Asp Val Val Cys Gly Gly Phe Ala Met Pro Ile Arg Glu Asn Lys
130 135 140
Ala Gln Glu Ile Tyr Ile Val Cys Ser Gly Glu Met Met Ala Met Tyr
145 150 155 160
Ala Ala Asn Asn Ile Ser Lys Gly Ile Val Lys Tyr Ala Asn Ser Gly
165 170 175
Ser Val Arg Leu Gly Gly Leu Ile Cys Asn Ser Arg Asn Thr Asp Arg
180 185 190
Glu Asp Glu Leu Ile Ile Ala Leu Ala Asn Lys Leu Gly Thr Gln Met
195 200 205
Ile His Phe Val Pro Arg Asp Asn Val Val Gln Arg Ala Glu Ile Arg
210 215 220
Arg Met Thr Val Ile Glu Tyr Asp Pro Lys Ala Lys Gln Ala Asp Glu
225 230 235 240
Tyr Arg Ala Leu Ala Arg Lys Val Val Asp Asn Lys Leu Leu Val Ile
245 250 255
Pro Asn Pro Ile Thr Met Asp Glu Leu Glu Glu Leu Leu Met Glu Phe
260 265 270
Gly Ile Met Glu Val Glu Asp Glu Ser Ile Val Gly Lys Thr Ala Glu
275 280 285
Glu Val
290
<210> 225
<211> 15
<212> PRT
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> X
<222> (12)..(12)
<223> любая аминокислота
<400> 225
Tyr Gly Lys Gly Gly Ile Gly Lys Ser Thr Thr Xaa Gln Asn Thr
1. 5 10 15
<210> 226
<211> 9
<212> PRT
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 226
Ile His Gly Cys Asp Pro Lys Ala Asp
1. 5
<210> 227
<211> 17
<212> PRT
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 227
Cys Val Glu Ser Gly Gly Pro Glu Pro Gly Val Gly Cys Ala Gly Arg
1. 5 10 15
Gly
<210> 228
<211> 14
<212> PRT
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 228
Asp Val Leu Gly Asp Val Val Cys Gly Gly Phe Ala Met Pro
1. 5 10
<210> 229
<211> 15
<212> PRT
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> X
<222> (9)..(9)
<223> любая аминокислота
<400> 229
Val Ala Ser Gly Glu Met Met Ala Xaa Tyr Ala Ala Asn Asn Ile
1. 5 10 15
<210> 230
<211> 5
<212> PRT
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 230
Gln Ser Gly Val Arg
1. 5
<210> 231
<211> 7
<212> PRT
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> X
<222> (5)..(5)
<223> любая аминокислота
<400> 231
Cys Asn Ser Arg Xaa Val Asp
1. 5
<210> 232
<211> 92
<212> PRT
<213> Azotobacter vinelandii
<400> 232
Met Ala Leu Lys Ile Val Glu Ser Cys Val Asn Cys Trp Ala Cys Val
1. 5 10 15
Asp Val Cys Pro Ser Glu Ala Ile Ser Leu Ala Gly Pro His Phe Glu
20 25 30
Ile Ser Ala Ser Lys Cys Thr Glu Cys Asp Gly Asp Tyr Ala Glu Lys
35 40 45
Gln Cys Ala Ser Ile Cys Pro Val Glu Gly Ala Ile Leu Leu Ala Asp
50 55 60
Gly Thr Pro Ala Asn Pro Pro Gly Ser Leu Thr Gly Ile Pro Pro Glu
65 70 75 80
Arg Leu Ala Glu Ala Met Arg Glu Ile Gln Ala Arg
85 90
<210> 233
<211> 157
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетический слитой полипептид
<400> 233
Met Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro
1. 5 10 15
Ser Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser
20 25 30
Asp Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val
35 40 45
Val Arg Asn Arg Gly Gly Ala Leu Lys Ile Val Glu Ser Cys Val Asn
50 55 60
Cys Trp Ala Cys Val Asp Val Cys Pro Ser Glu Ala Ile Ser Leu Ala
65 70 75 80
Gly Pro His Phe Glu Ile Ser Ala Ser Lys Cys Thr Glu Cys Asp Gly
85 90 95
Asp Tyr Ala Glu Lys Gln Cys Ala Ser Ile Cys Pro Val Glu Gly Ala
100 105 110
Ile Leu Leu Ala Asp Gly Thr Pro Ala Asn Pro Pro Gly Ser Leu Thr
115 120 125
Gly Ile Pro Pro Glu Arg Leu Ala Glu Ala Met Arg Glu Ile Gln Ala
130 135 140
Arg Ala Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala
145 150 155
<210> 234
<211> 156
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетический слитой полипептид
<400> 234
Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro Ser
1. 5 10 15
Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser Asp
20 25 30
Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val Val
35 40 45
Arg Asn Arg Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Gly Gly
50 55 60
Ala Leu Lys Ile Val Glu Ser Cys Val Asn Cys Trp Ala Cys Val Asp
65 70 75 80
Val Cys Pro Ser Glu Ala Ile Ser Leu Ala Gly Pro His Phe Glu Ile
85 90 95
Ser Ala Ser Lys Cys Thr Glu Cys Asp Gly Asp Tyr Ala Glu Lys Gln
100 105 110
Cys Ala Ser Ile Cys Pro Val Glu Gly Ala Ile Leu Leu Ala Asp Gly
115 120 125
Thr Pro Ala Asn Pro Pro Gly Ser Leu Thr Gly Ile Pro Pro Glu Arg
130 135 140
Leu Ala Glu Ala Met Arg Glu Ile Gln Ala Arg Ala
145 150 155
<210> 235
<211> 156
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетический слитой полипептид
<400> 235
Met Ala Met Ala Val Phe Arg Arg Glu Ala Ala Ala Leu Leu Pro Ser
1. 5 10 15
Ile Ala Ala Arg Pro Ile Ala Ala Ala Ala Ala Ala Ala Ser Ser Asp
20 25 30
Gln Glu Glu Gly Leu Leu Ala Ala Ala Ala Ala Ala Ala Ala Val Val
35 40 45
Arg Asn Arg Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Gly Gly
50 55 60
Ala Leu Lys Ile Val Glu Ser Cys Val Asn Cys Trp Ala Cys Val Asp
65 70 75 80
Val Cys Pro Ser Glu Ala Ile Ser Leu Ala Gly Pro His Phe Glu Ile
85 90 95
Ser Ala Ser Lys Cys Thr Glu Cys Asp Gly Asp Tyr Ala Glu Lys Gln
100 105 110
Cys Ala Ser Ile Cys Pro Val Glu Gly Ala Ile Leu Leu Ala Asp Gly
115 120 125
Thr Pro Ala Asn Pro Pro Gly Ser Leu Thr Gly Ile Pro Pro Glu Arg
130 135 140
Leu Ala Glu Ala Met Arg Glu Ile Gln Ala Arg Ala
145 150 155
<210> 236
<211> 104
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетический слитой полипептид
<400> 236
Met Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Gly Gly Ala Leu Lys Ile
1. 5 10 15
Val Glu Ser Cys Val Asn Cys Trp Ala Cys Val Asp Val Cys Pro Ser
20 25 30
Glu Ala Ile Ser Leu Ala Gly Pro His Phe Glu Ile Ser Ala Ser Lys
35 40 45
Cys Thr Glu Cys Asp Gly Asp Tyr Ala Glu Lys Gln Cys Ala Ser Ile
50 55 60
Cys Pro Val Glu Gly Ala Ile Leu Leu Ala Asp Gly Thr Pro Ala Asn
65 70 75 80
Pro Pro Gly Ser Leu Thr Gly Ile Pro Pro Glu Arg Leu Ala Glu Ala
85 90 95
Met Arg Glu Ile Gln Ala Arg Ala
100
<210> 237
<211> 448
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетический слитой полипептид
<400> 237
Met Ala Met Ala Val Phe Arg Arg Glu Gly Arg Arg Leu Leu Pro Ser
1. 5 10 15
Ile Ala Ala Arg Pro Ile Ala Ala Ile Arg Ser Pro Leu Ser Ser Asp
20 25 30
Gln Glu Glu Gly Leu Leu Gly Val Arg Ser Ile Ser Thr Gln Val Val
35 40 45
Arg Asn Arg Gly Gly Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Gly Gly
50 55 60
Met Ala Ser Val Ile Ile Asp Asp Thr Thr Leu Arg Asp Gly Glu Gln
65 70 75 80
Ser Ala Gly Val Ala Phe Asn Ala Asp Glu Lys Ile Ala Ile Ala Arg
85 90 95
Ala Leu Ala Glu Leu Gly Val Pro Glu Leu Glu Ile Gly Ile Pro Ser
100 105 110
Met Gly Glu Glu Glu Arg Glu Val Met His Ala Ile Ala Gly Leu Gly
115 120 125
Leu Ser Ser Arg Leu Leu Ala Trp Cys Arg Leu Cys Asp Val Asp Leu
130 135 140
Ala Ala Ala Arg Ser Thr Gly Val Thr Met Val Asp Leu Ser Leu Pro
145 150 155 160
Val Ser Asp Leu Met Leu His His Lys Leu Asn Arg Asp Arg Asp Trp
165 170 175
Ala Leu Arg Glu Val Ala Arg Leu Val Gly Glu Ala Arg Met Ala Gly
180 185 190
Leu Glu Val Cys Leu Gly Cys Glu Asp Ala Ser Arg Ala Asp Leu Glu
195 200 205
Phe Val Val Gln Val Gly Glu Val Ala Gln Ala Ala Gly Ala Arg Arg
210 215 220
Leu Arg Phe Ala Asp Thr Val Gly Val Met Glu Pro Phe Gly Met Leu
225 230 235 240
Asp Arg Phe Arg Phe Leu Ser Arg Arg Leu Asp Met Glu Leu Glu Val
245 250 255
His Ala His Asp Asp Phe Gly Leu Ala Thr Ala Asn Thr Leu Ala Ala
260 265 270
Val Met Gly Gly Ala Thr His Ile Asn Thr Thr Val Asn Gly Leu Gly
275 280 285
Glu Arg Ala Gly Asn Ala Ala Leu Glu Glu Cys Val Leu Ala Leu Lys
290 295 300
Asn Leu His Gly Ile Asp Thr Gly Ile Asp Thr Arg Gly Ile Pro Ala
305 310 315 320
Ile Ser Ala Leu Val Glu Arg Ala Ser Gly Arg Gln Val Ala Trp Gln
325 330 335
Lys Ser Val Val Gly Ala Gly Val Phe Thr His Glu Ala Gly Ile His
340 345 350
Val Asp Gly Leu Leu Lys His Arg Arg Asn Tyr Glu Gly Leu Asn Pro
355 360 365
Asp Glu Leu Gly Arg Ser His Ser Leu Val Leu Gly Lys His Ser Gly
370 375 380
Ala His Met Val Arg Asn Thr Tyr Arg Asp Leu Gly Ile Glu Leu Ala
385 390 395 400
Asp Trp Gln Ser Gln Ala Leu Leu Gly Arg Ile Arg Ala Phe Ser Thr
405 410 415
Arg Thr Lys Arg Ser Pro Gln Pro Ala Glu Leu Gln Asp Phe Tyr Arg
420 425 430
Gln Leu Cys Glu Gln Gly Asn Pro Glu Leu Ala Ala Gly Gly Met Ala
435 440 445
<210> 238
<211> 243
<212> PRT
<213> Azotobacter vinelandii
<400> 238
Met Val Thr Pro Val Asn Met Ser Arg Glu Thr Ala Leu Arg Ile Ala
1. 5 10 15
Leu Ala Ala Arg Ala Leu Pro Gly Thr Thr Val Gly Gln Leu Leu Glu
20 25 30
Ile Leu His Gln Arg Ile Glu Gly Pro Leu Thr Glu Glu Ser Leu Gln
35 40 45
Gly Val Ser Val Thr Asp Leu Lys Ile Gly Leu Ala Gly Ser Glu Glu
50 55 60
Asp Val Asp Met Leu Asp Thr Pro Met Ser Ala Leu Lys Asp Ala Val
65 70 75 80
Arg Ile Leu Trp Gly Glu Ala Glu Val Asp Ser Leu Pro Gln Pro Val
85 90 95
Lys Leu Glu Arg Val Pro Glu Gly Ser Ile Arg Val Ala Ile Ala Ser
100 105 110
Asn Asn Gly Glu Gln Leu Asp Gly His Phe Gly Ser Cys Leu Arg Phe
115 120 125
Leu Val Tyr Gln Val Ser Ala Lys Asp Ala Ser Leu Val Asp Ile Arg
130 135 140
Ser Thr Leu Asp Val Ala Leu Ala Glu Asp Lys Asn Ala Trp Arg Val
145 150 155 160
Glu Gln Ile Gln Asp Cys Gln Val Leu Tyr Val Val Ser Ile Gly Gly
165 170 175
Pro Ala Ala Ala Lys Val Val Arg Ala Gly Ile His Pro Leu Lys Lys
180 185 190
Pro Lys Gly Cys Ala Ala Gln Glu Ala Ile Ala Glu Leu Gln Thr Val
195 200 205
Met Ala Gly Ser Pro Pro Pro Trp Leu Ala Lys Leu Val Gly Val Ser
210 215 220
Ala Glu Glu Arg Val Arg Phe Ser Val Ser Asp Asp Glu Asp Glu Ala
225 230 235 240
Ala Arg Ala
<210> 239
<211> 4
<212> PRT
<213> Искусственная последовательность
<220>
<223> С-концевая аминокислотная последовательность
<400> 239
Asp Leu Ile Arg
1
<210> 240
<211> 4
<212> PRT
<213> Искусственная последовательность
<220>
<223> С-концевая аминокислотная последовательность
<400> 240
Asp Val Val Arg
1
<210> 241
<211> 4
<212> PRT
<213> Искусственная последовательность
<220>
<223> С-концевая аминокислотная последовательность
<400> 241
Asp Ile Ile Arg
1
<210> 242
<211> 4
<212> PRT
<213> Искусственная последовательность
<220>
<223> С-концевая аминокислотная последовательность
<400> 242
Asp Leu Thr Arg
1
<210> 243
<211> 4
<212> PRT
<213> Искусственная последовательность
<220>
<223> С-концевая аминокислотная последовательность
<400> 243
Ile Asn Val Trp
1
<210> 244
<211> 4
<212> PRT
<213> Искусственная последовательность
<220>
<223> С-концевая аминокислотная последовательность
<400> 244
Leu Asn Val Trp
1
<210> 245
<211> 4
<212> PRT
<213> Искусственная последовательность
<220>
<223> С-концевая аминокислотная последовательность
<400> 245
Leu Asn Thr Trp
1
<210> 246
<211> 4
<212> PRT
<213> Искусственная последовательность
<220>
<223> С-концевая аминокислотная последовательность
<400> 246
Leu Asn Met Trp
1
<210> 247
<211> 4
<212> PRT
<213> Искусственная последовательность
<220>
<223> С-концевая аминокислотная последовательность
<400> 247
Leu Ala Met Trp
1
<210> 248
<211> 4
<212> PRT
<213> Искусственная последовательность
<220>
<223> С-концевая аминокислотная последовательность
<400> 248
Leu Ser Val Trp
1
1/269
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ЭКСПРЕССИЯ ПОЛИПЕПТИДОВ НИТРОГЕНАЗЫ В РАСТИТЕЛЬНЫХ КЛЕТКАХ | 2018 |
|
RU2809244C2 |
| СЛИТЫЕ СЕРПИНОВЫЕ ПОЛИПЕПТИДЫ И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2019 |
|
RU2728861C1 |
| СЛИТЫЕ СЕРПИНОВЫЕ ПОЛИПЕПТИДЫ И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2015 |
|
RU2746550C2 |
| КОНСТРУКЦИИ СЛИТОГО БЕЛКА ДЛЯ ЗАБОЛЕВАНИЯ, СВЯЗАННОГО С КОМПЛЕМЕНТОМ | 2019 |
|
RU2824402C2 |
| СЛИТЫЕ СЕРПИНОВЫЕ ПОЛИПЕПТИДЫ И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2012 |
|
RU2698655C2 |
| СЛИТЫЙ БЕЛОК GDF15 ДЛИТЕЛЬНОГО ДЕЙСТВИЯ И СОДЕРЖАЩАЯ ЕГО ФАРМАЦЕВТИЧЕСКАЯ КОМПОЗИЦИЯ | 2020 |
|
RU2836280C1 |
| СЛИТЫЙ БЕЛОК ИЗ БЕЛКА DCTN1 С БЕЛКОМ RET | 2018 |
|
RU2813996C2 |
| ПОЛИВАЛЕТНЫЕ И ПОЛИСПЕЦИФИЧНЫЕ GITR-СВЯЗЫВАЮЩИЕ СЛИТЫЕ БЕЛКИ | 2016 |
|
RU2753439C2 |
| СЛИТЫЙ ПОЛИПЕПТИД С ПРОТИВОРАКОВОЙ АКТИВНОСТЬЮ | 2016 |
|
RU2754466C2 |
| МУЛЬТИВАЛЕНТНЫЕ PD-L1-СВЯЗЫВАЮЩИЕ СОЕДИНЕНИЯ ДЛЯ ЛЕЧЕНИЯ ЗЛОКАЧЕСТВЕННЫХ НОВООБРАЗОВАНИЙ | 2020 |
|
RU2816646C2 |

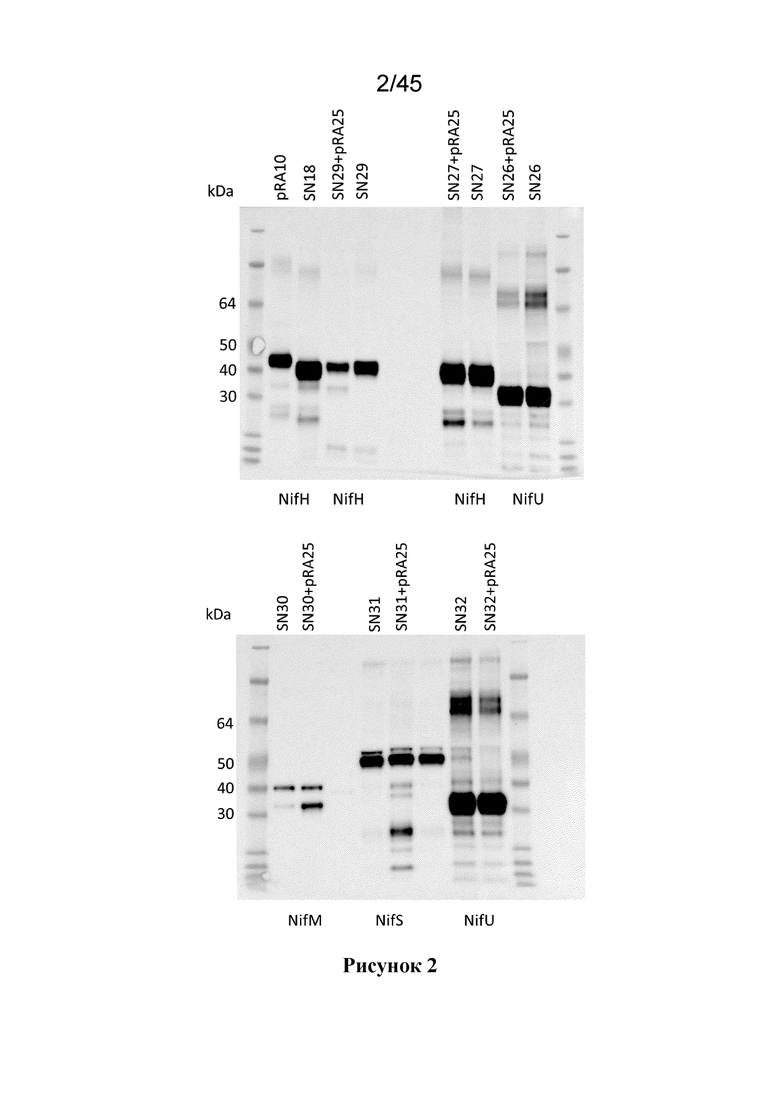
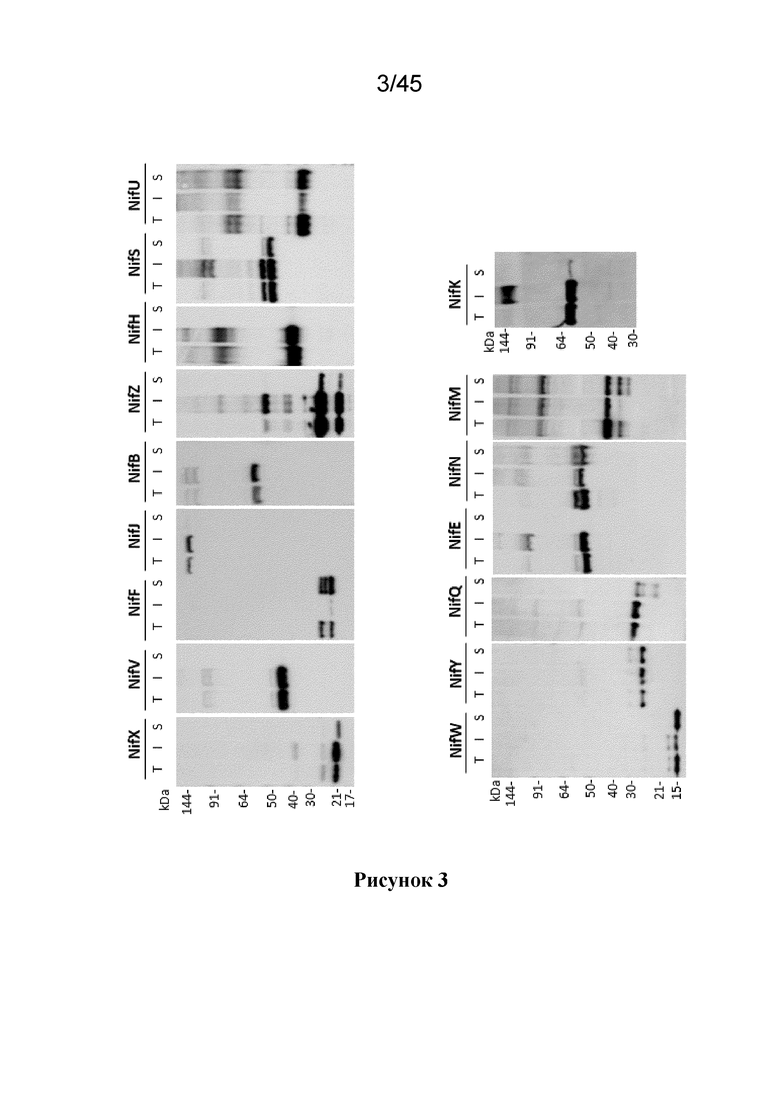
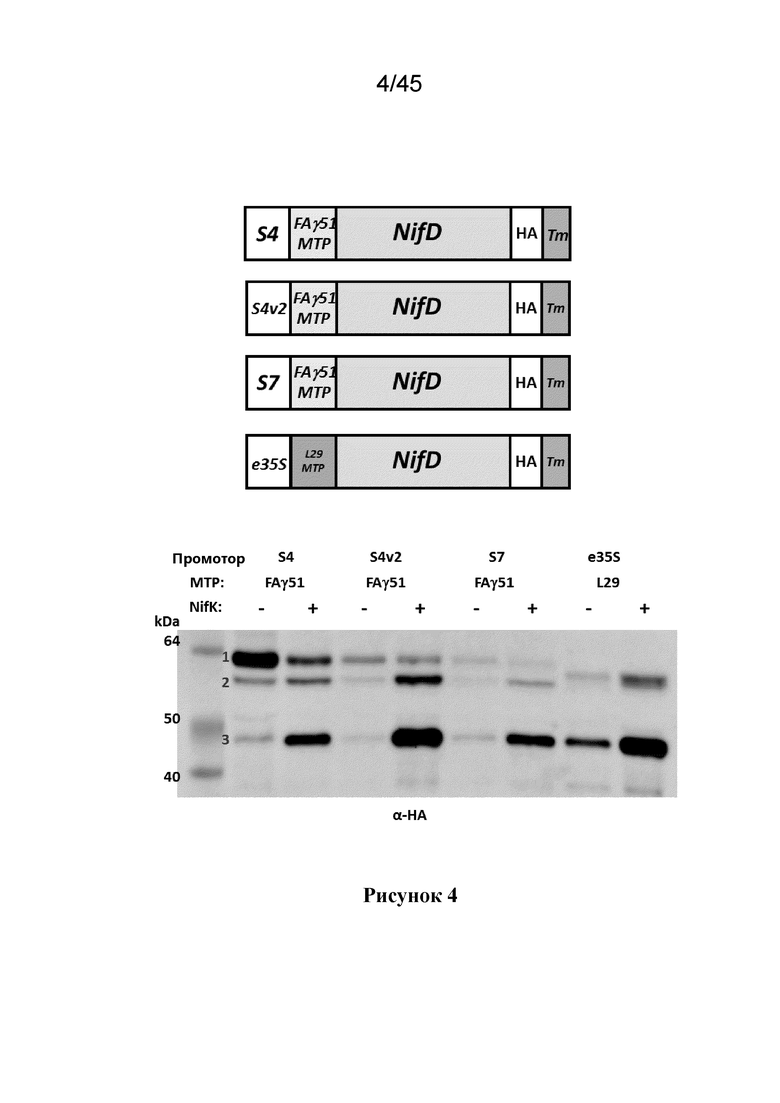

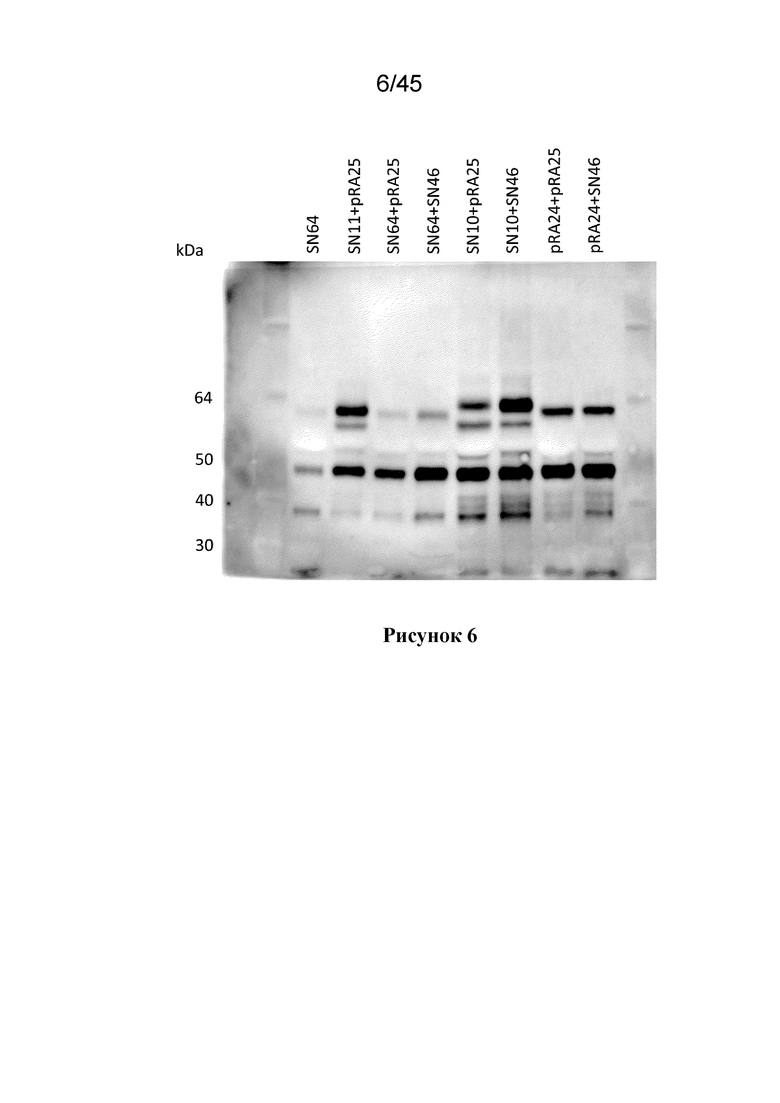

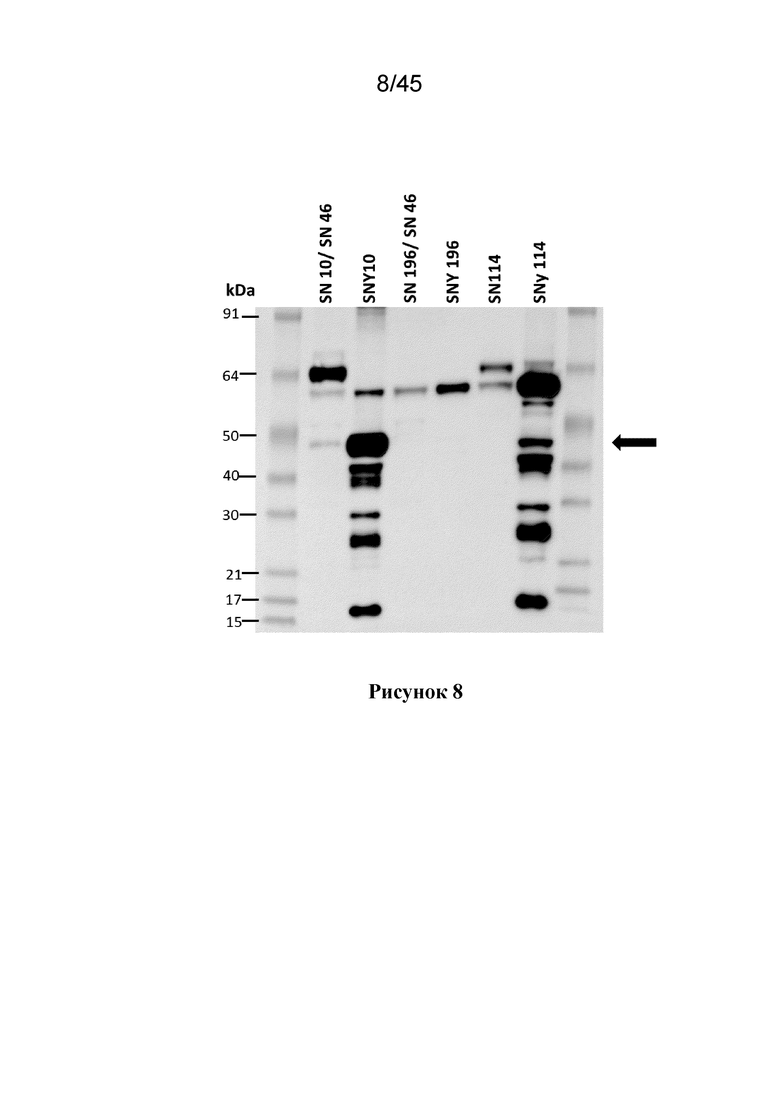
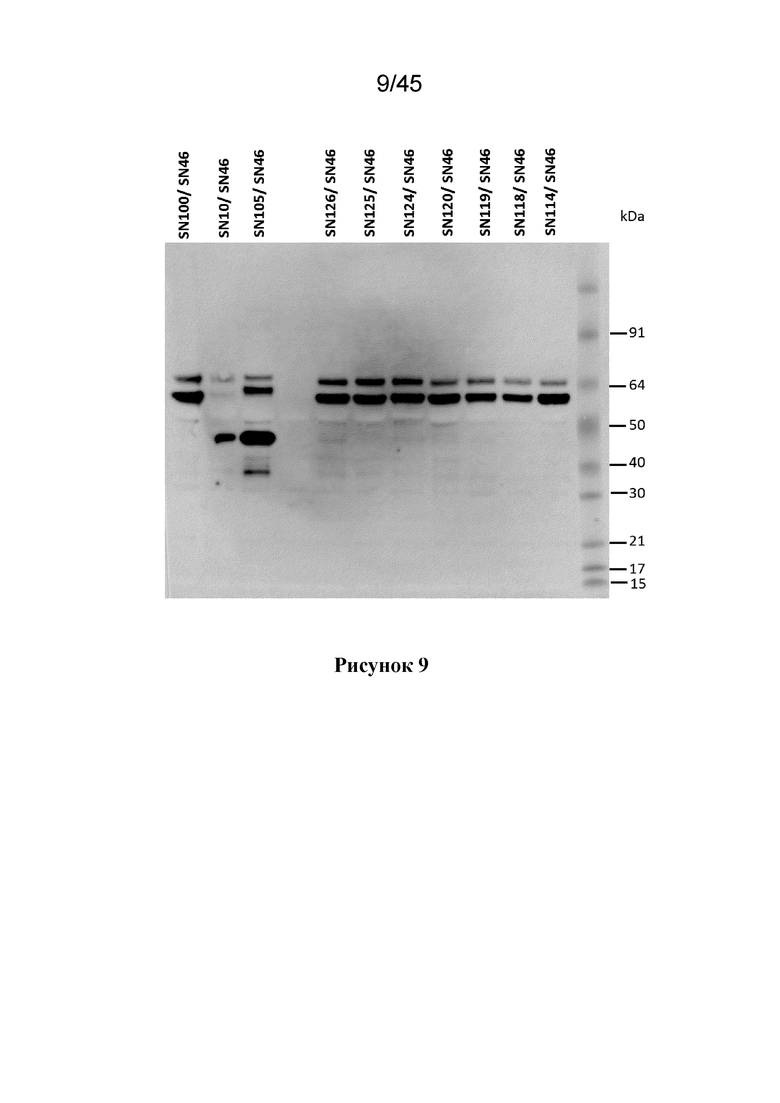


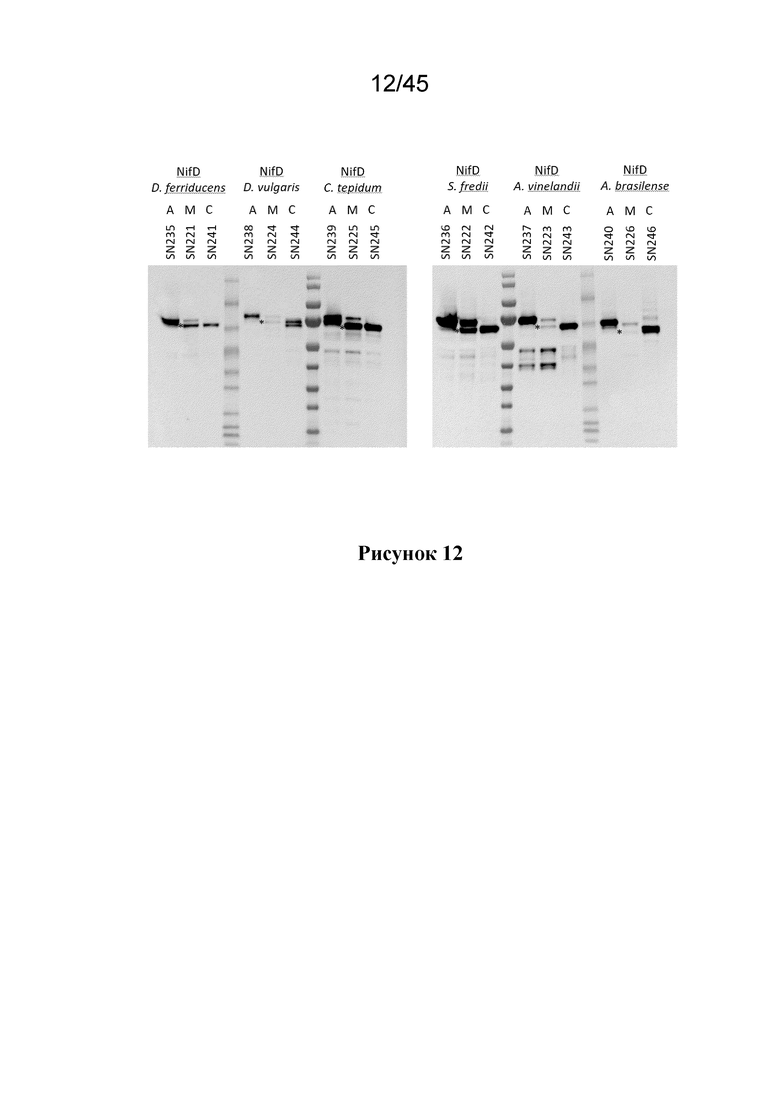
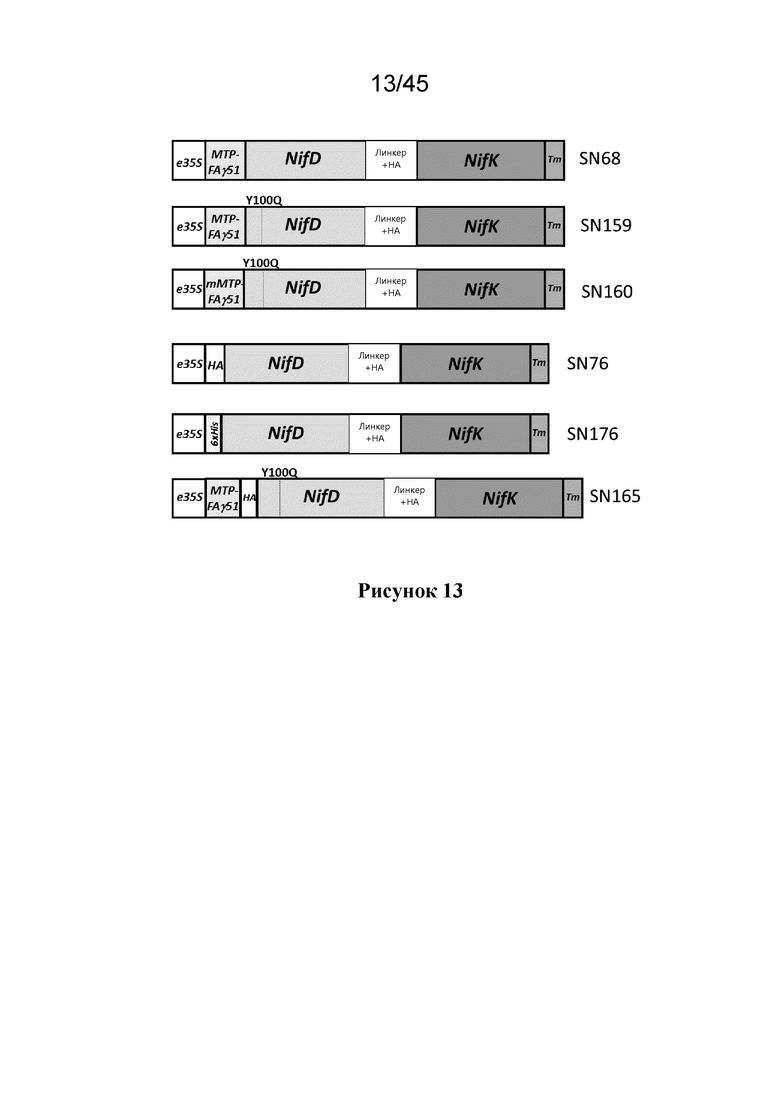
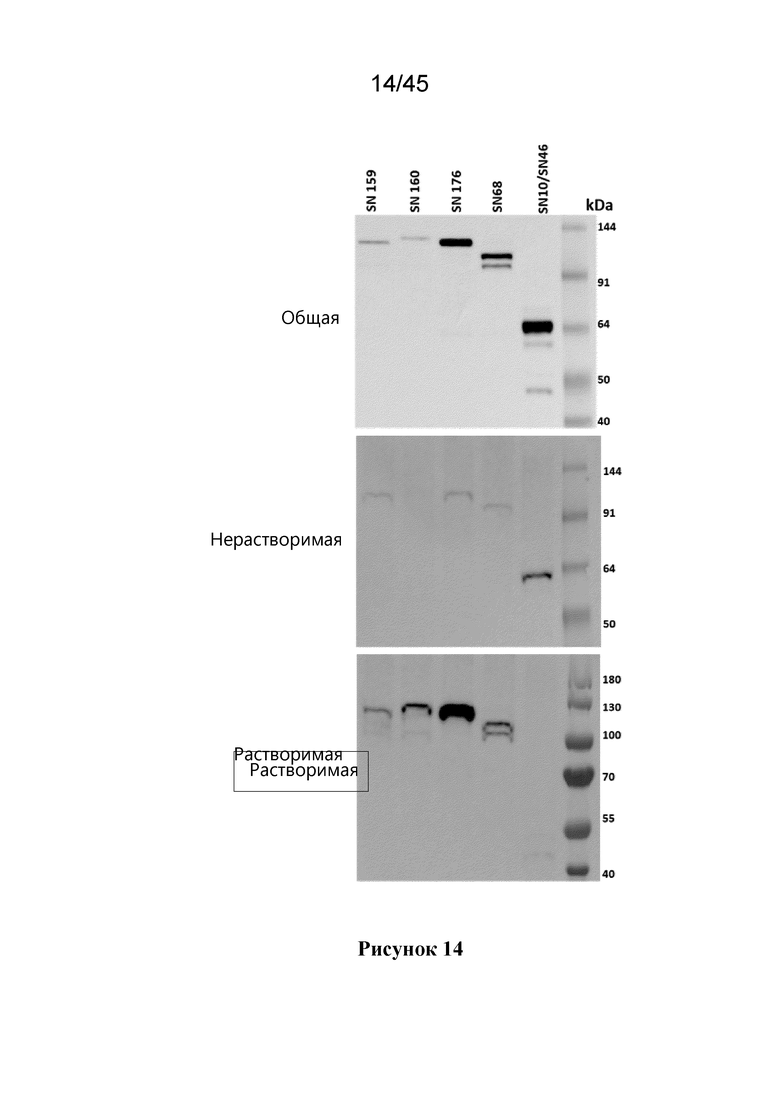
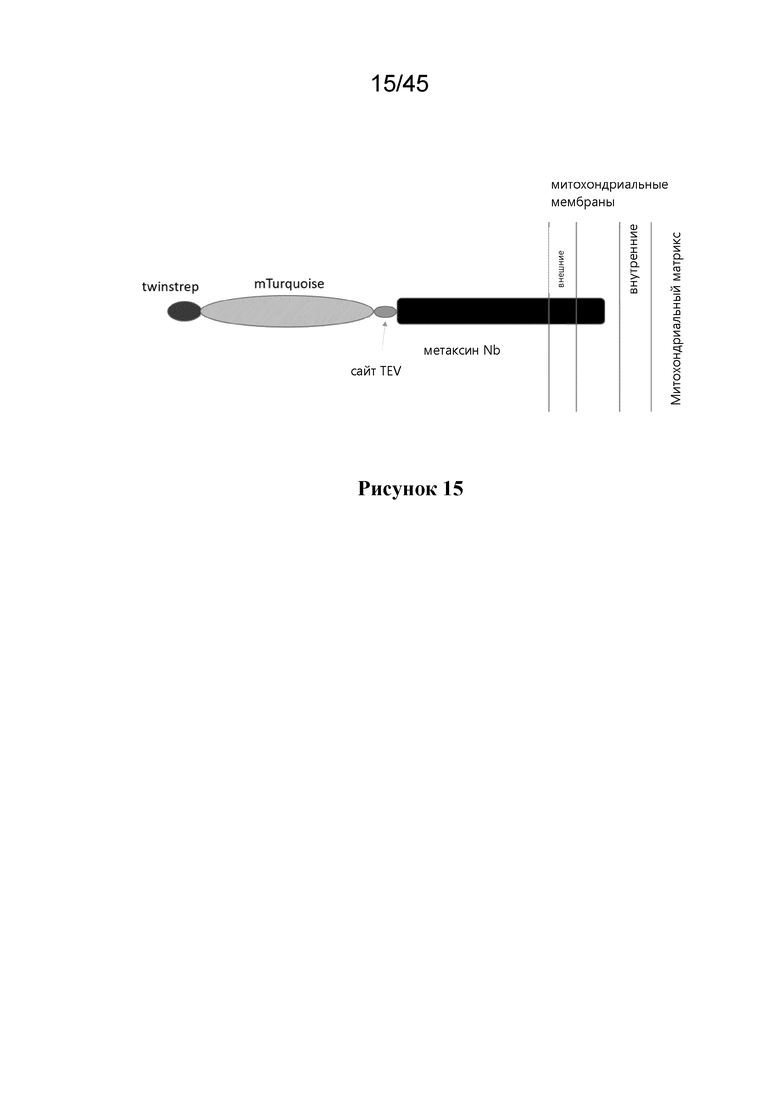
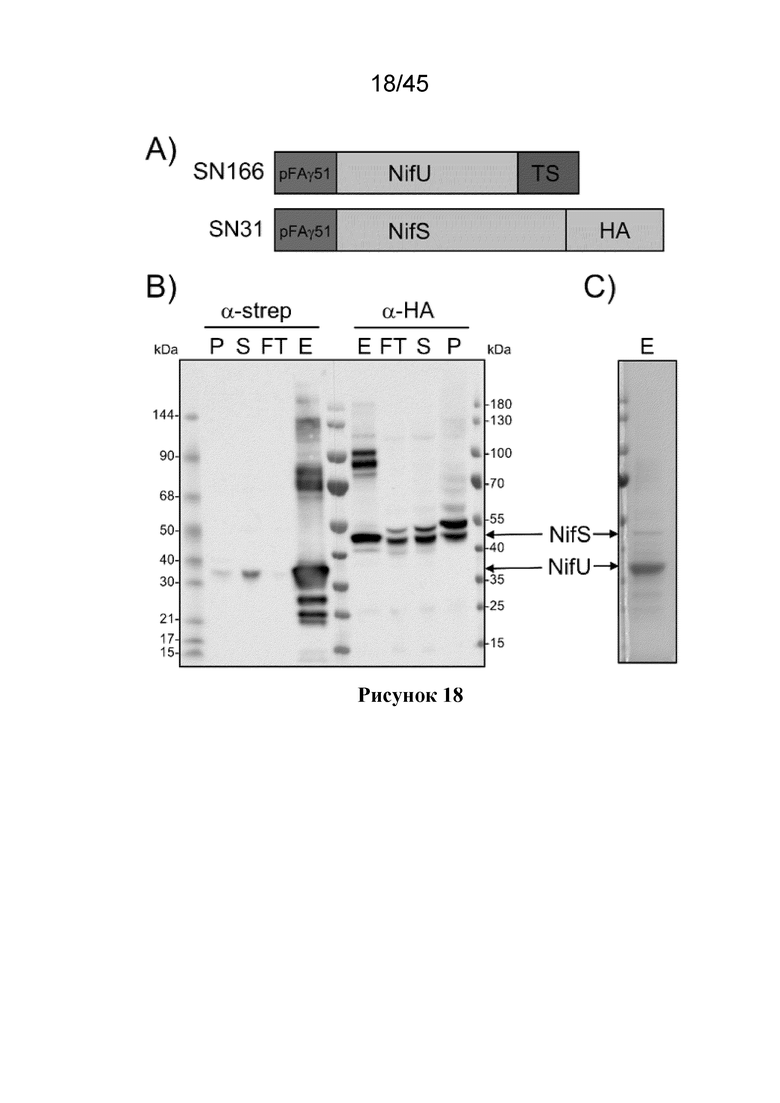
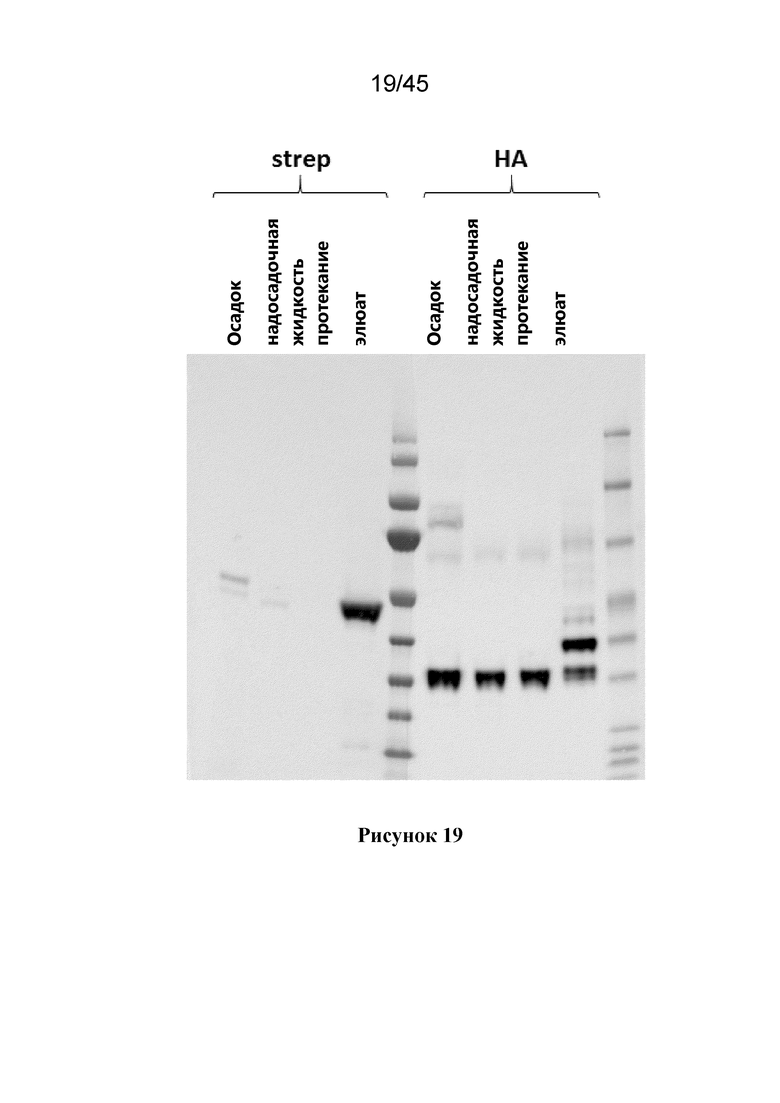



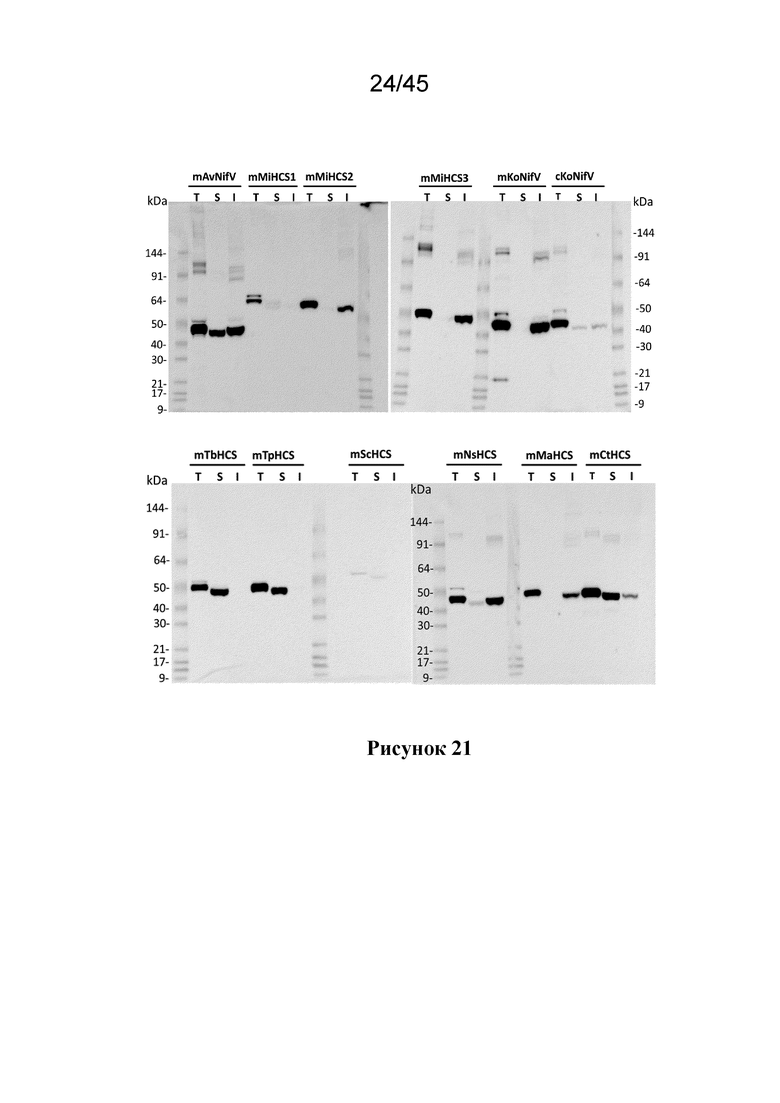
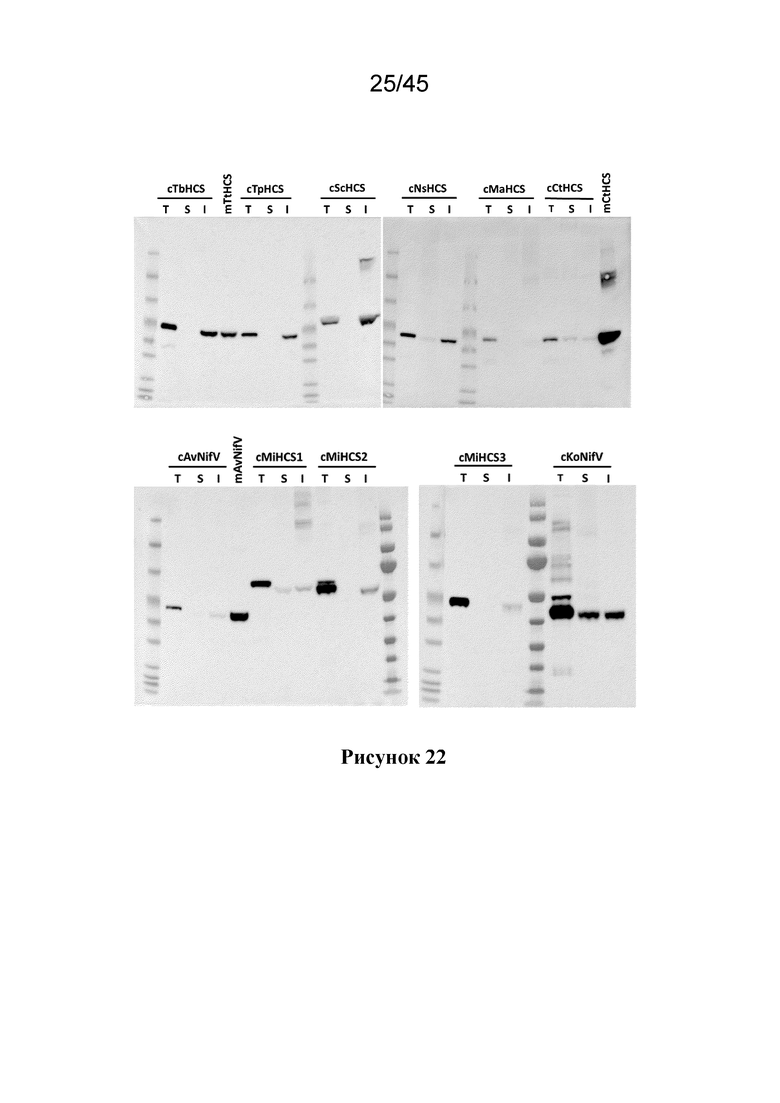
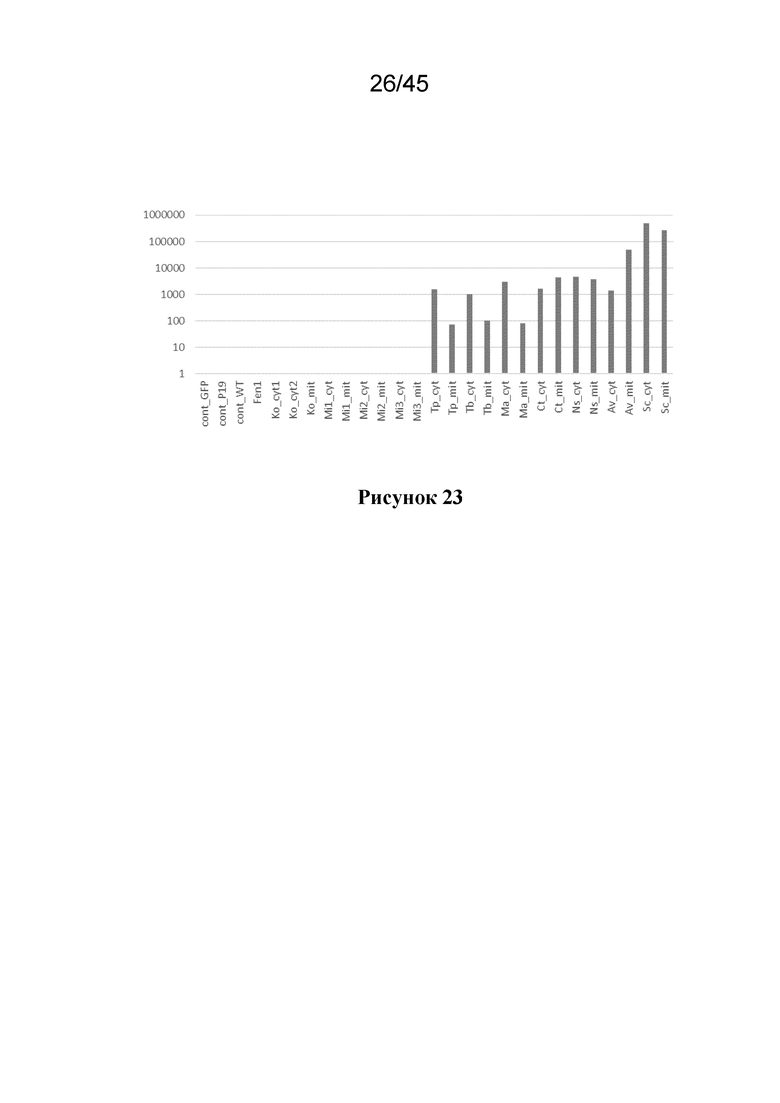
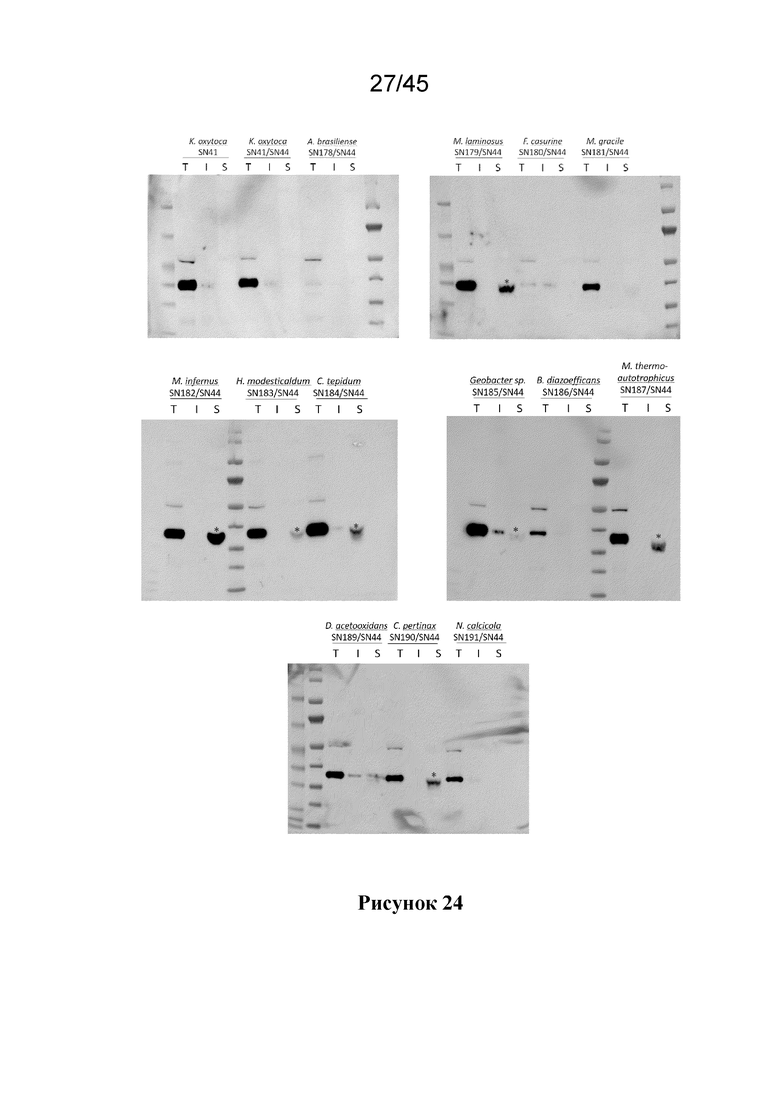


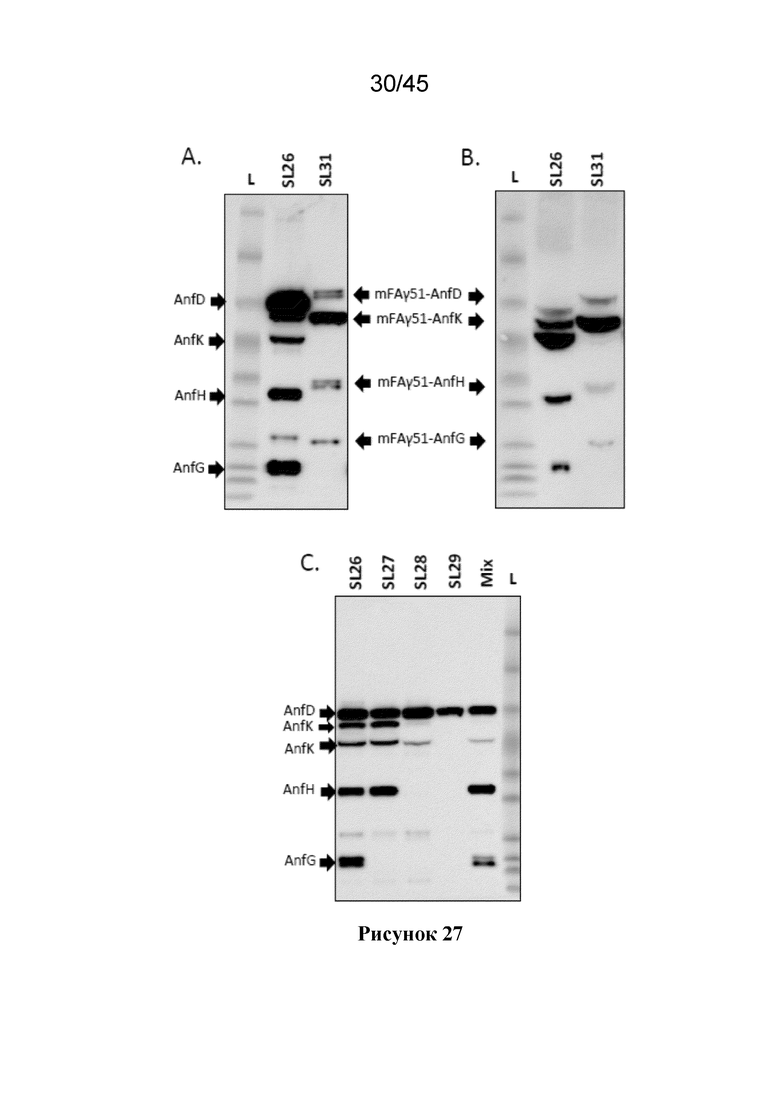
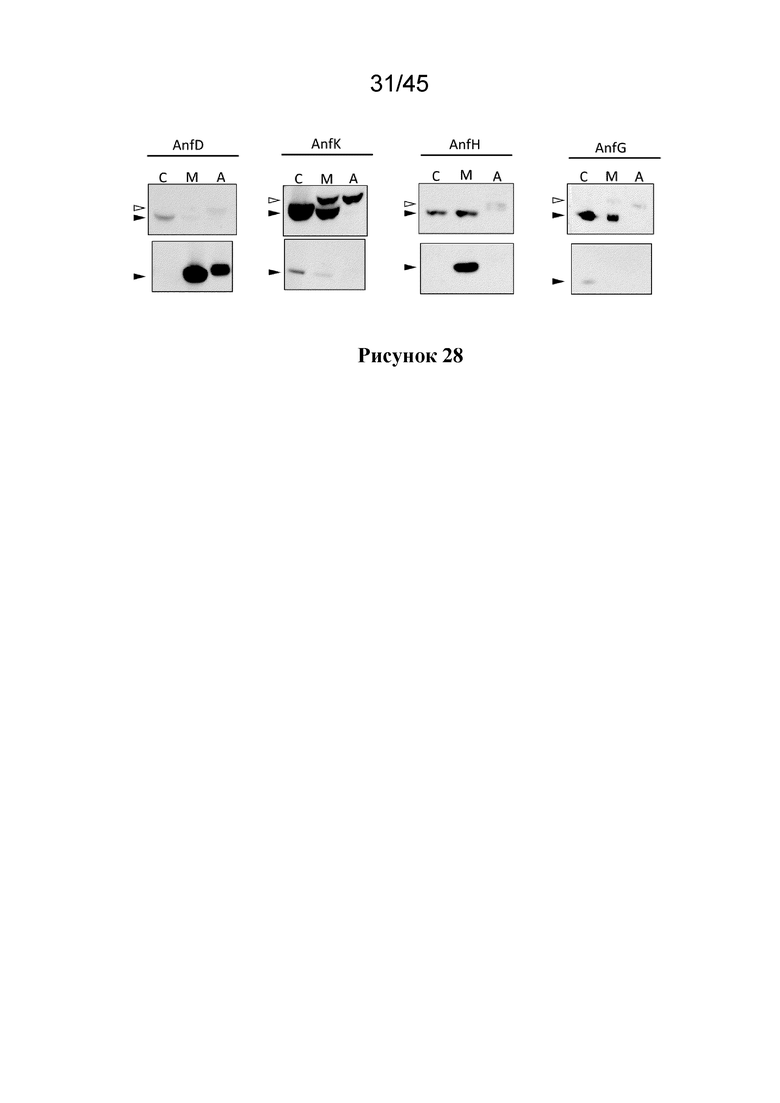

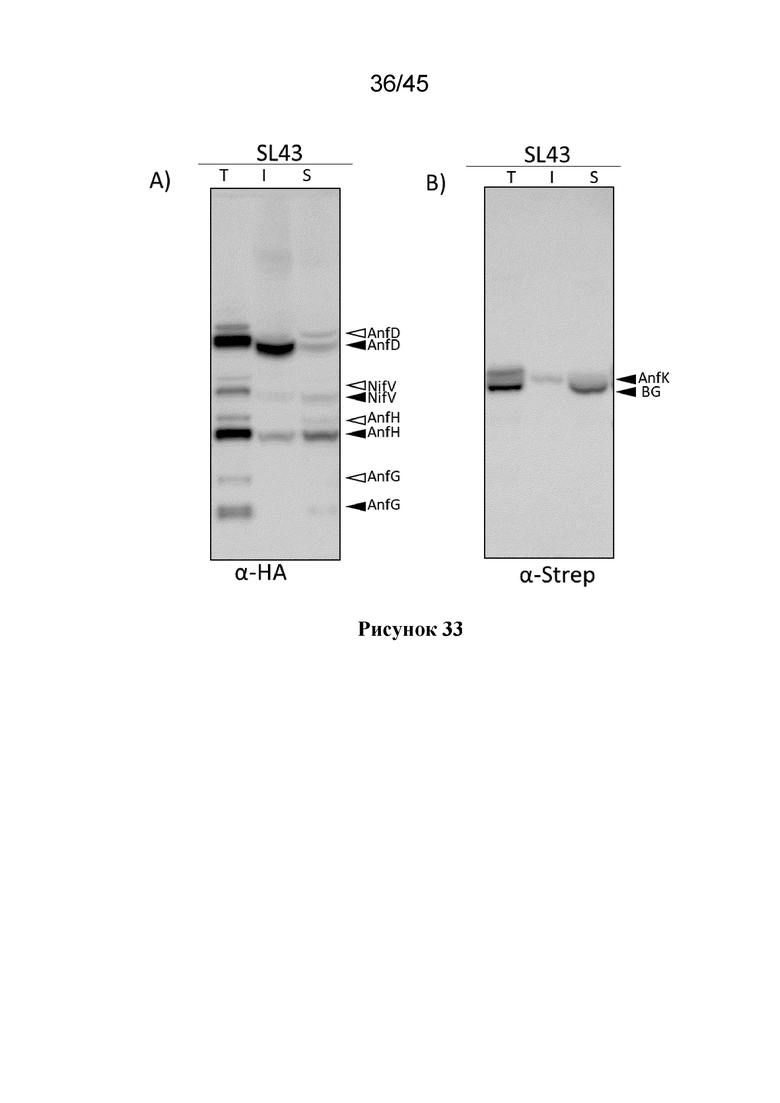
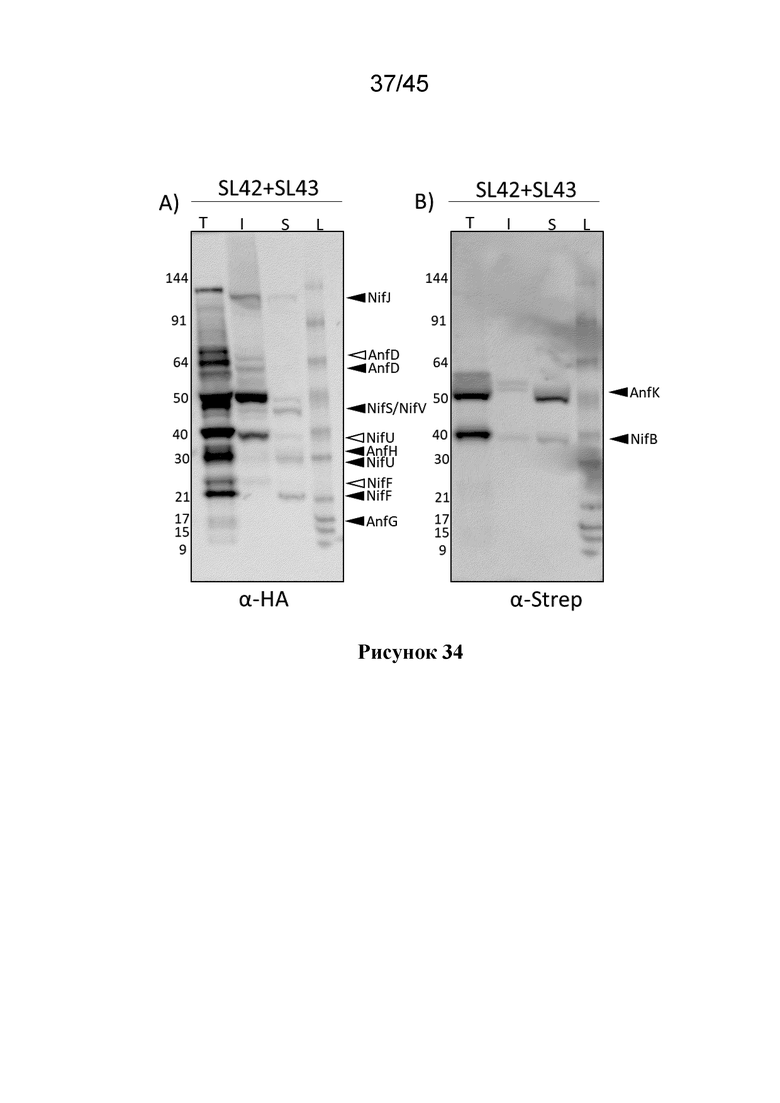
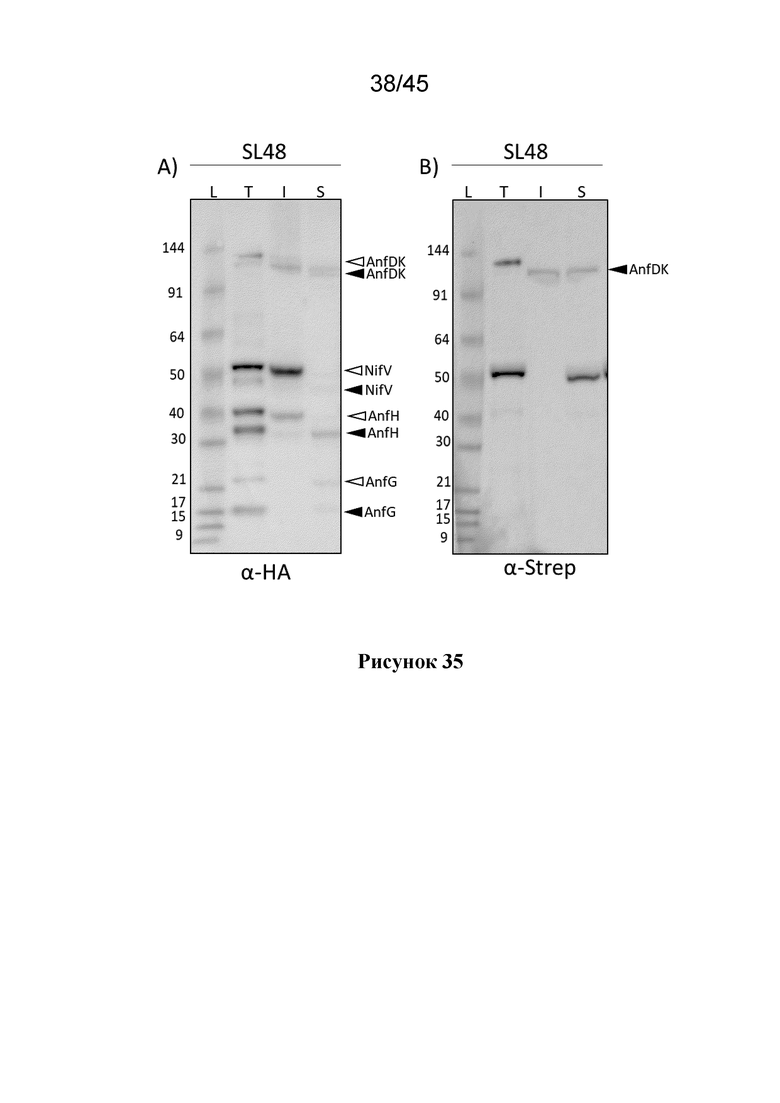
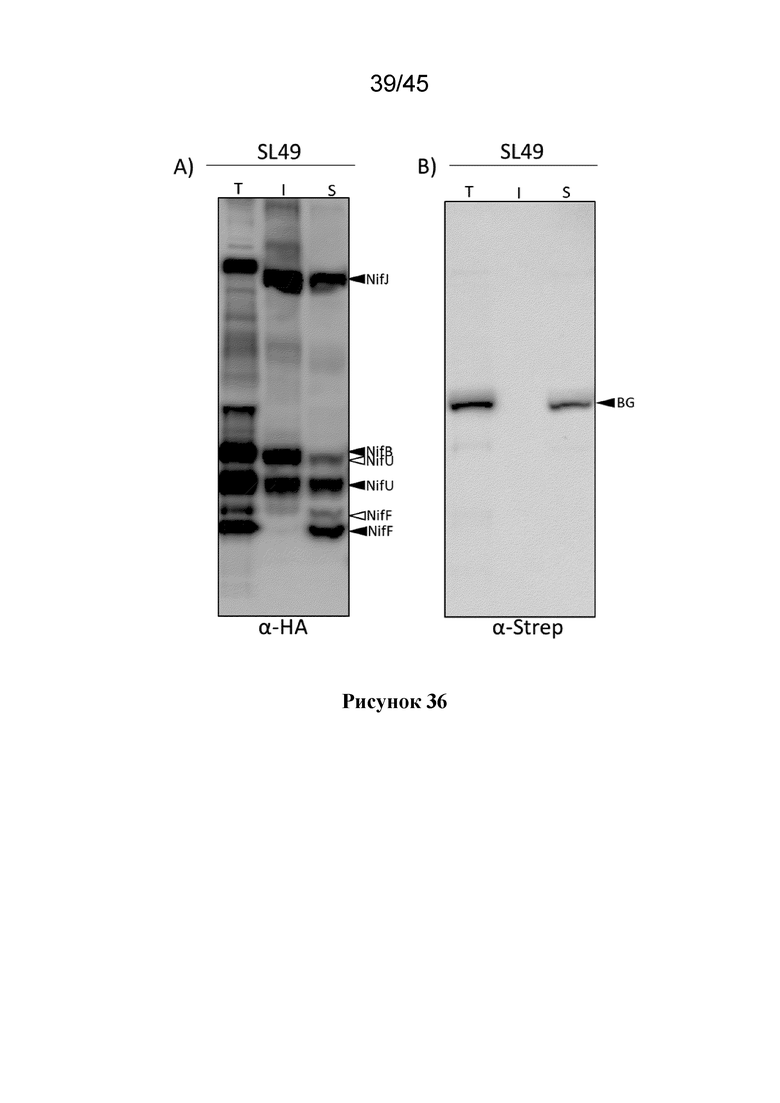
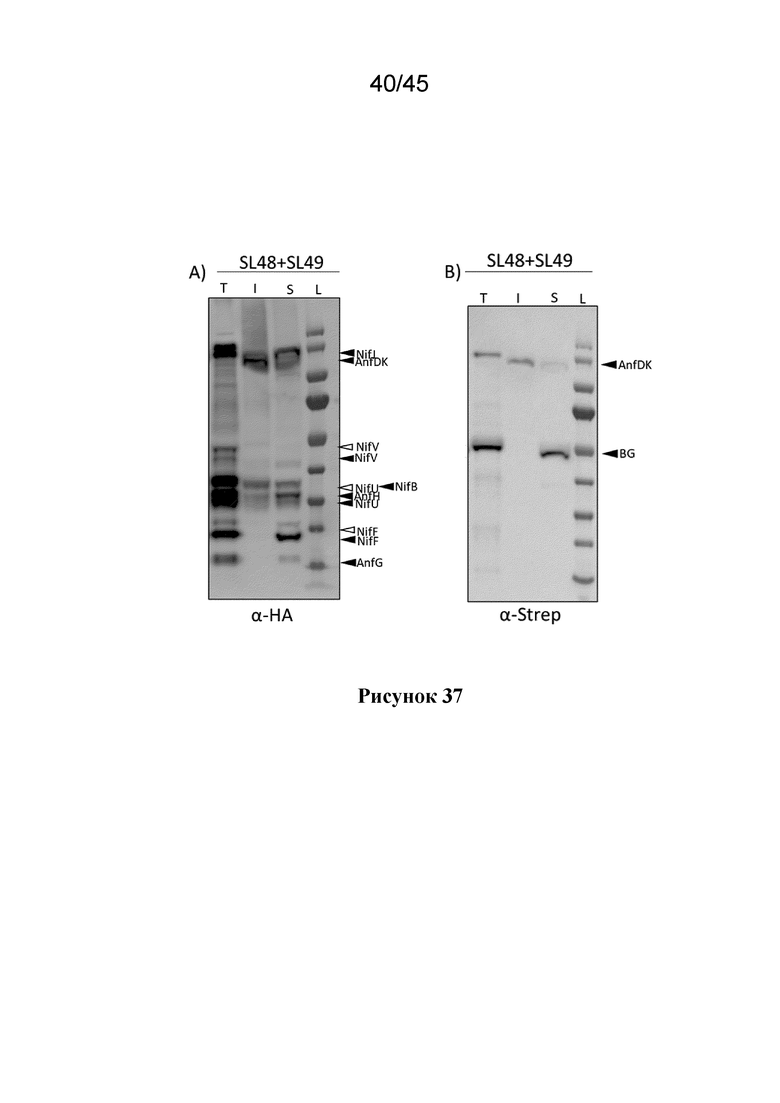
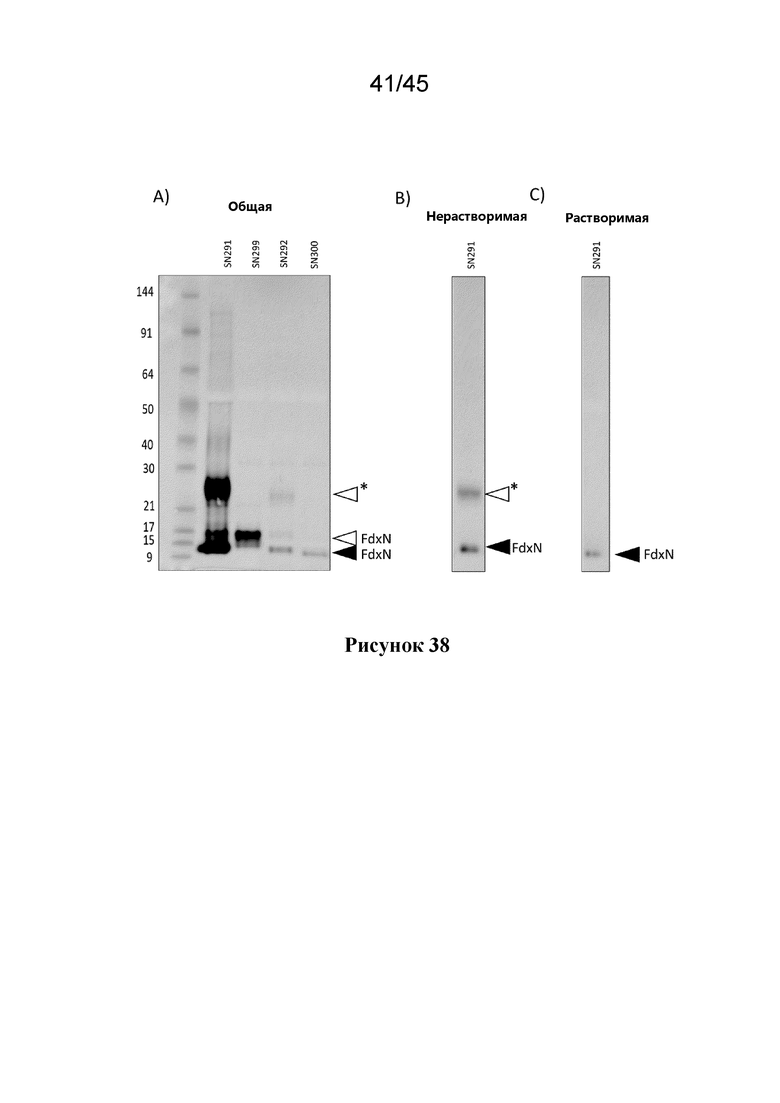
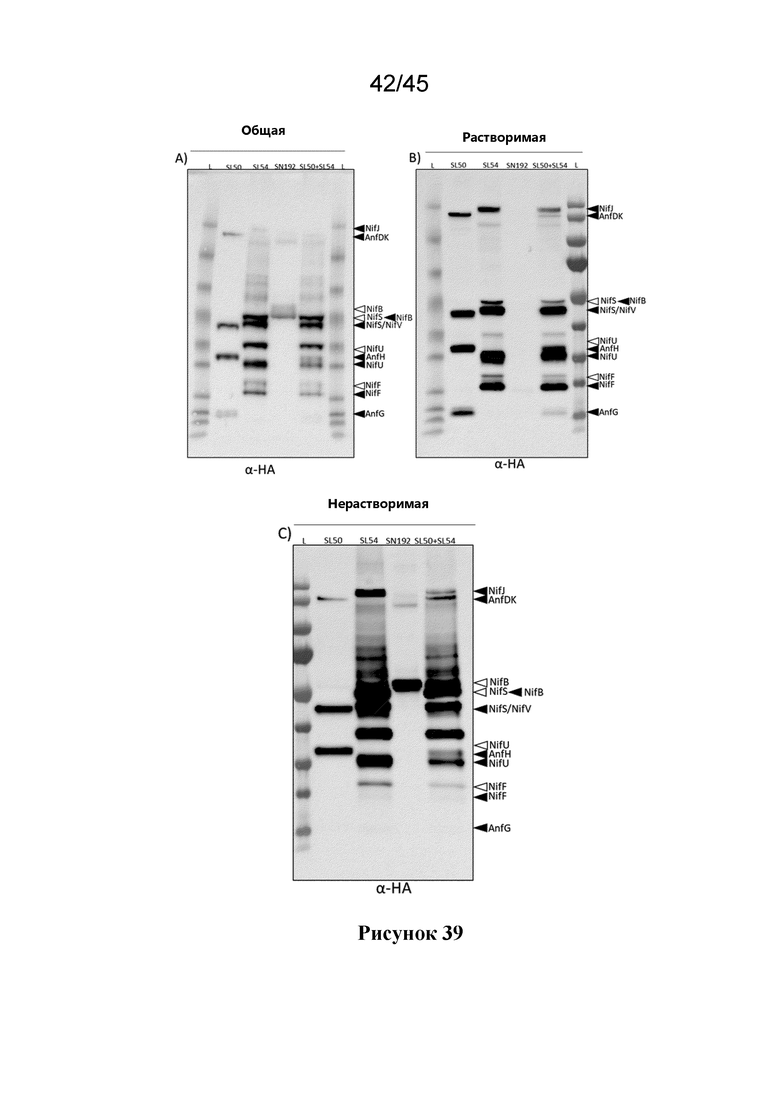

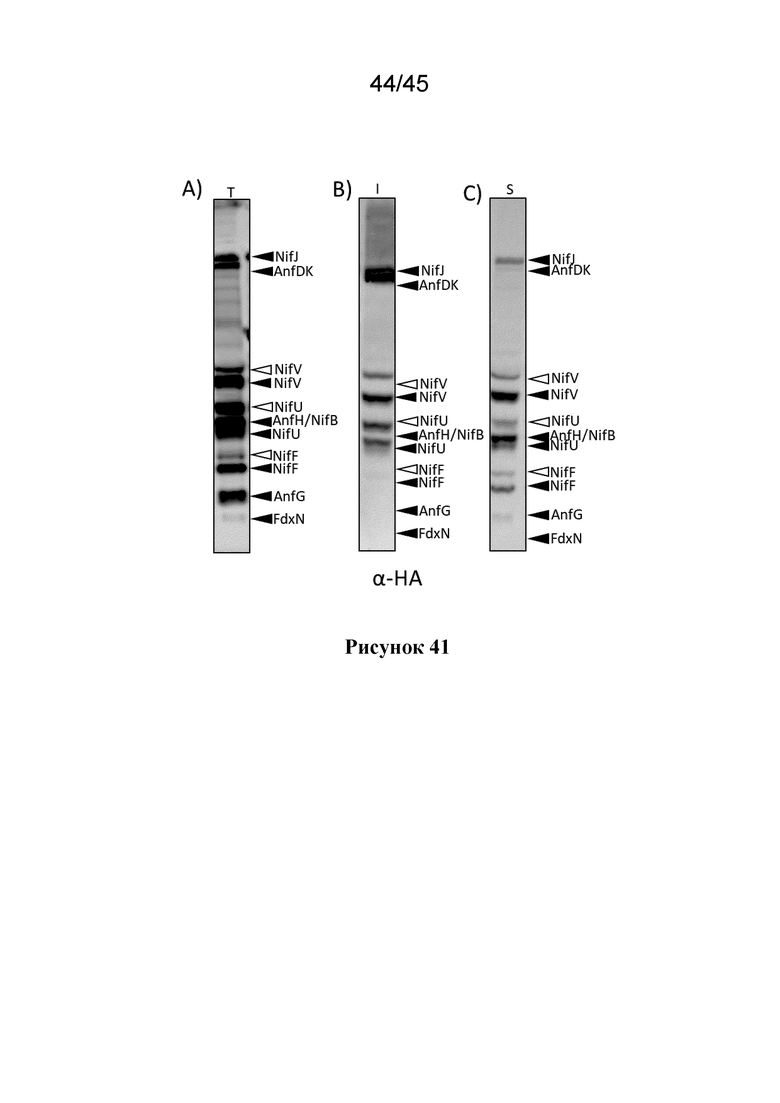
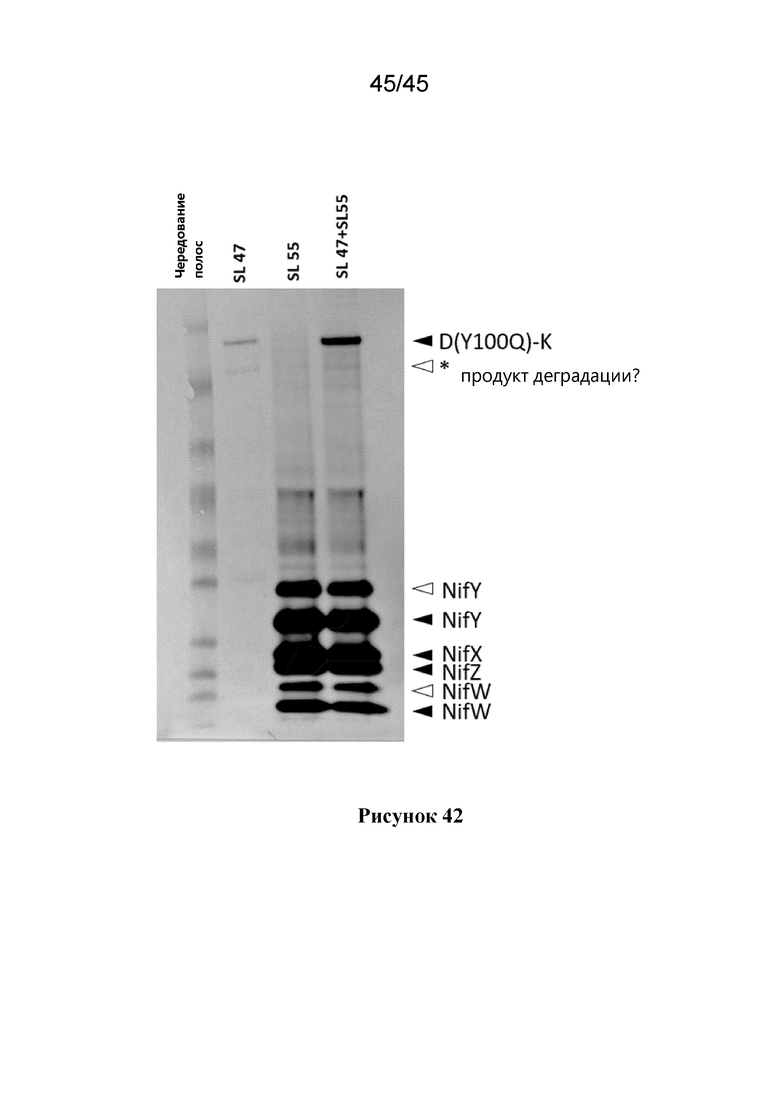
Изобретение относится к области биохимии, в частности к растительной клетке для экспрессии полипептида NifD. Также раскрыты растение или его часть, пищевой продукт, содержащие указанный полипептид; слитой полипептид NifD; комбинация слитых полипептидов NifD. Раскрыты применение указанного полинуклеотида для продуцирования трансгенной растительной клетки; способ получения указанного продукта; способ кормления животного указанным продуктом. Изобретение позволяет эффективно получать полипептид NifD. 14 н. и 33 з.п. ф-лы, 42 ил., 23 табл., 24 пр.
1. Растительная клетка для экспрессии полипептида NifD, содержащая экзогенный полинуклеотид, который кодирует полипептид NifD, который: (а) устойчив к расщеплению протеазой на сайте в аминокислотной последовательности, соответствующей аминокислотам 97-100 из SEQ ID NO:18; и (b) содержит аминокислотную последовательность, отличную от RRNY (SEQ ID NO:101) в позициях, соответствующих аминокислотам 97-100 из SEQ ID NO:18, где экзогенный полинуклеотид содержит промотор, который функционально связан с нуклеотидной последовательностью, которая кодирует полипептид NifD и которая экспрессирует указанную нуклеотидную последовательность в растительной клетке.
2. Растительная клетка по п. 1, отличающаяся тем, что полипептид NifD представляет собой слитой полипептид, который содержит митохондриальный нацеливающий пептид (MTP), при этом MTP, предпочтительно, трансляционно слит с N-концом полипептида NifD.
3. Растительная клетка по п. 2, отличающаяся тем, что слитой полипептид NifD расщепляется внутри MTP митохондриальной процессинговой протеазой (MPP) с продуцированием MPP-расщепленного продукта, причем MPP-расщепленный продукт, содержит: (i) в своей N-концевой области либо C-концевой пептид из MTP (пептид scar) либо (ii) не содержит C-концевой пептид из MTP.
4. Растительная клетка по п. 3, отличающаяся тем, что MPP-расщепленный продукт, по меньшей мере, частично растворим в митохондриях растительной клетки.
5. Растительная клетка по любому из пп. 1-4, содержащая экзогенный полинуклеотид, кодирующий полипептид NifK (NK), отличающаяся тем, что экзогенный полинуклеотид, кодирующий NK, содержит промотор, который функционально связан с нуклеотидной последовательностью, кодирующей NK, и который экспрессирует указанную нуклеотидную последовательность в растительной клетке, где полипептид NifD (ND) имеет С-конец, а NK имеет N-конец, и где (i) NK представляет собой слитой полипептид NifK, который включает MTP, или (ii) ND и NK трансляционно слитые в виде слитого полипептида NifD-линкер-NifK, который содержит олигопептидный линкер, где олигопептидный линкер трансляционно слит с C-концом ND и N-концом NK.
6. Растительная клетка по п. 5, содержащая экзогенный полинуклеотид, который кодирует слитой полипептид NifD-линкер-NifK, отличающаяся тем, что олигопептидный линкер имеет длину в 8-50 остатков, предпочтительно в 16-50 остатков, более предпочтительно примерно в 26 или примерно в 30 остатков или наиболее предпочтительно в 30 остатков.
7. Растительная клетка по п. 5 или 6, отличающаяся тем, что слитой полипептид NifK или слитой полипептид NifD-линкер-NifK имеет С-концевую аминокислотную последовательность, которая аналогична С-концевой аминокислотной последовательности полипептида NifK дикого типа.
8. Растительная клетка по п. 7, отличающаяся тем, что слитой полипептид NifK или слитой полипептид NifD-линкер-NifK имеет аминокислотную последовательность, в которой последние четыре аминокислоты из последовательности аналогичны последним четырем аминокислотам полипептида NifK дикого типа.
9. Растительная клетка по любому из пп. 6-8, отличающаяся тем, что слитой полипептид NifD-линкер-NifK расщепляется внутри MTP с продуцированием MPP-расщепленного продукта, где MPP-расщепленный продукт слитого полипептида NifD-линкер-NifK, по меньшей мере, частично растворим в митохондриях растительной клетки.
10. Растительная клетка по любому из пп. 1-9, отличающаяся тем, что слитой полипептид NifD или слитой полипептид NifD-линкер-NifK содержит отличную от тирозина (Y) аминокислоту в позиции, соответствующей аминокислоте 100 в SEQ ID NO:18, и предпочтительно содержит глутамин (Q) или лизин (K) в позиции, соответствующей аминокислоте 100 в SEQ ID NO:18.
11. Растительная клетка по любому из пп. 1-10, содержащая экзогенный полинуклеотид, который кодирует слитой полипептид NifH, отличающаяся тем, что экзогенный полинуклеотид, который кодирует слитой полипептид NifH, содержит промотор, который функционально связан с нуклеотидной последовательностью, кодирующей слитой полипептид NifH, и который экспрессирует указанную нуклеотидную последовательность в растительной клетке, где слитой полипептид NifH содержит MTP, предпочтительно, где MTP находится на N-конце слитого полипептида NifH.
12. Растительная клетка по п. 11, отличающаяся тем, что слитой полипептид NifH расщепляется внутри MTP с продуцированием MPP-расщепленного продукта, причем MPP-расщепленный продукт содержит: (i) в своей N-концевой области либо C-концевой пептид из MTP (пептид scar) либо (ii) не содержит C-концевой пептид из MTP.
13. Растительная клетка по п. 12, отличающаяся тем, что MPP-расщепленный продукт слитого полипептида NifH, по меньшей мере, частично растворим в митохондриях растительной клетки.
14. Растительная клетка по любому из пп. 1-13, дополнительно содержащая один или несколько или все:
(i) экзогенный полинуклеотид, который кодирует полипептид NifV (NV), где экзогенный полинуклеотид содержит промотор, который функционально связан с нуклеотидной последовательностью, кодирующей NV и экспрессирующей указанную нуклеотидную последовательность в растительной клетке, где NV продуцирует гомоцитрат в растительной клетке и где, необязательно, NV содержит MTP,
(ii) экзогенный полинуклеотид, который кодирует полипептид NifM (NM), где экзогенный полинуклеотид, кодирующий NM, содержит промотор, который функционально связан с нуклеотидной последовательностью, кодирующей NM и экспрессирующей указанную нуклеотидную последовательность в растительной клетке, и где, необязательно, NM содержит MTP, и
(iii) экзогенные полинуклеотиды, которые кодируют слитой полипептид NifS (NS) и слитой полипептид NifU (NU), где каждый экзогенный полинуклеотид содержит промотор, который функционально связан с нуклеотидной последовательностью, которая кодирует один из слитых полипептидов Nif и которая экспрессирует нуклеотидную последовательность в растительной клетке, и где каждый из NS и NU содержит MTP.
15. Растительная клетка по п. 14, отличающаяся тем, что один или несколько или все NV, NM, NS и NU расщепляются внутри MTP с независимым продуцированием MPP-расщепленного продукта, причем MPP-расщепленный продукт в каждом случае содержит: (i) в своей N-концевой области либо C-концевой пептид из MTP (пептид scar) либо (ii) не содержит C-концевой пептид из MTP.
16. Растительная клетка по любому из пп. 1-15, содержащая экзогенные полинуклеотиды, которые кодируют, по меньшей мере, 8 или, по меньшей мере, 9 слитых полипептидов Nif, отличающаяся тем, что каждый экзогенный полинуклеотид содержит промотор, который функционально связан с нуклеотидной последовательностью, которая кодирует один из слитых полипептидов Nif, и которая экспрессируют нуклеотидную последовательность в растительной клетке, в которой каждый слитой полипептид Nif независимо содержит MTP, при этом слитые полипептиды Nif содержат: (i) слитые полипептиды NifH, NifB, NifF, NifJ, NifS, NifU и NifV; и (ii) либо слитой полипептид NifD и слитой полипептид NifK; или (iii) слитой полипептид NifD-линкер-NifK, который содержит: последовательность NifD, имеющую С-конец; олигопептидный линкер; и последовательность NifK, имеющую N-конец; при этом олигопептидный линкер трансляционно слит с С-концом последовательности NifD и N-концом последовательности NifK; при этом MPP-расщепленные продукты, по меньшей мере, слитых полипептидов NifH, NifF, NifS и NifU каждый, по меньшей мере, частично растворим в митохондриях растительной клетки; при этом MPP-расщепленные продукты слитых полипептидов NifD и NifK из (ii), если они присутствуют в растительной клетке, по меньшей мере частично растворимы в митохондриях растительной клетки, или MPP-расщепленный продукт слитого полипептида NifD-линкер-NifK из (iii), если он присутствует в растительной клетке, по меньшей мере частично растворим в митохондриях растительной клетки, и при этом слитой полипептид NifV и/или его MPP-расщепленный продукт продуцирует гомоцитрат в растительной клетке и, по меньшей мере, частично растворим в митохондриях растительной клетки.
17. Растительная клетка по любому из пп. 2-16, отличающаяся тем, что каждый MTP независимо расщепляется в растительной клетке с эффективностью, составляющей, по меньшей мере, 50%, и/или где каждый MPP-расщепленный продукт независимо присутствует в растительной клетке на более высоком уровне, чем соответствующий нерасщепленный слитой полипептид Nif, предпочтительно в соотношении, составляющем более чем 1:1, более чем 2:1 или более чем 3:1.
18. Растительная клетка по любому из пп. 2-17, отличающаяся тем, что каждый слитой полипептид Nif, по меньшей мере, частично расщеплен в своей последовательности MTP в растительной клетке с независимым продуцированием MPP-расщепленного продукта, причем каждый MPP-расщепленный продукт содержит пептид (пептид scar) длиной от 1 до 45 аминокислот, предпочтительно от 1 до 20 аминокислот, более предпочтительно от 1 до 11 аминокислот или от 11 до 20 аминокислот, полученных из последовательности MTP, где пептид scar трансляционно слит в N-концевой области MPP-расщепленного продукта.
19. Растительная клетка по любому из пп. 11-18, отличающаяся тем, что экзогенные полинуклеотиды кодируют: (i) слитой полипептид NifH, который представляет собой слитой полипептид AnfH; и либо (ii) слитой полипептид NifD, который представляет собой слитой полипептид AnfD и слитой полипептид NifK, который представляет собой слитой полипептид AnfK; или (iii) слитой полипептид NifD-линкер-NifK, который представляет собой слитой полипептид AnfD-линкер-AnfK; причем растительная клетка дополнительно содержит (iv) экзогенный полинуклеотид, который кодирует слитой полипептид AnfG, который содержит MTP, где экзогенный полинуклеотид, который кодирует слитой полипептид AnfG, содержит промотор, который функционально связан с нуклеотидной последовательностью, кодирующей слитой полипептид AnfG и которая экспрессирует указанную нуклеотидную последовательность в растительной клетке, и где MPP-расщепленный продукт слитого полипептида AnfG, по меньшей мере, частично растворим в митохондриях растительной клетки.
20. Растительная клетка по любому из пп. 1-19, отличающаяся тем, что один или несколько или предпочтительно все экзогенные полинуклеотиды интегрированы в ядерный геном растительной клетки и/или экспрессируются в ядре растительной клетки.
21. Растение для экспрессии полипептида NifD, содержащее растительную клетку по любому из пп. 1-20.
22. Растение по п. 21, которое представляет собой злаковое растение, предпочтительно, растение пшеницу, рис, кукурузу, тритикале, овес или ячмень.
23. Часть растения для экспрессии полипептида NifD, содержащая растительную клетку по любому из пп. 1-20.
24. Часть растения по п. 23, полученная из злакового растения, предпочтительно, растения пшеницы, риса, кукурузы, тритикале, овса или ячменя.
25. Слитой полипептид NifD для экспрессии в растительной клетке, где слитой полипептид NifD содержит митохондриальный нацеливающий пептид (MTP), трансляционно слитой с полипептидом NifD (ND), или его MPP-расщепленный продукт, который содержит ND, где расщепленный продукт содержит на его N-конце C-концевой пептид из MTP,
где слитой полипептид NifD или его расщепленный продукт: (а) устойчив к расщеплению протеазой на сайте в аминокислотной последовательности, соответствующей аминокислотам 97-100 из SEQ ID NO:18; и (b) содержит аминокислотную последовательность, отличную от RRNY (SEQ ID NO:101) в позициях, соответствующих аминокислотам 97-100 из SEQ ID NO:18.
26. Слитой полипептид NifD или расщепленный продукт по п. 25, содержащий отличную от тирозина (Y) аминокислоту в позиции, соответствующей аминокислоте 100 из SEQ ID NO:18, предпочтительно содержащий глутамин (Q) или лизин (K) в позиции, соответствующей аминокислоте 100 в SEQ ID NO:18.
27. Слитой полипептид NifD или расщепленный продукт по п. 25 или 26, который содержит олигопептидный линкер и полипептид NifK (NK), которые трансляционно слиты с ND в качестве слитого полипептида NifD-линкер-NifK, отличающийся тем, что ND содержит С-конец, а NK содержит N-конец, где олигопептидный линкер трансляционно слит с C-концом ND и N-концом NK.
28. Слитой полипептид NifD или его расщепленный продукт по любому из пп. 25-27, который, по меньшей мере, частично растворим в митохондриях растительной клетки, когда слитой полипептид NifD продуцируется в растительной клетке.
29. Слитой полипептид NifD или его расщепленный продукт по любому из пп. 25-28, который находится в растительной клетке, предпочтительно в митохондриях растительной клетки.
30. Слитой полипептид NifD или его расщепленный продукт по любому из пп. 26-29, отличающийся тем, что слитой полипептид NifD представляет собой слитой полипептид AnfD, NK представляет собой полипептид AnfK, а слитой полипептид NifD- линкер-NifK представляет собой слитой полипептид AnfD-линкер-AnfK.
31. Комбинация слитых полипептидов Nif для экспрессии в растительной клетке, включающая (i) слитой полипептид NifD или его расщепленного продукта по п. 25 или 26 и (ii) слитой полипептид NifK, который содержит MTP, трансляционно слитый с полипептидом NifK (NK), или его MPP-расщепленный продукт, отличающаяся тем, что слитой полипептид NifK или его MPP-расщепленный продукт, по меньшей мере, частично растворимы в митохондриях растительной клетки, когда слитой полипептид NifK или его MPP-расщепленный продукт продуцируется в растительной клетке.
32. Слитой полипептид или его расщепленный продукт по любому из пп. 25-30 или комбинация по п. 31, отличающиеся тем, что полипептид NifK имеет С-концевую аминокислотную последовательность, которая аналогична С-концевой аминокислотной последовательности полипептида NifK дикого типа.
33. Комбинация по п. 31 или 32, дополнительно содержащая слитой полипептид NifH, содержащий MTP, трансляционно слитой с полипептидом NifH (NH), или его расщепленный продукт, содержащий NH, отличающаяся тем, что слитой полипептид NifH и/или его расщепленный продукт, по меньшей мере, частично растворимы в митохондриях растительной клетки.
34. Комбинация по любому из пп. 31-33, отличающаяся тем, что ND представляет собой полипептид AnfD, NK представляет собой полипептид AnfK, а NH представляет собой полипептид AnfH.
35. Полинуклеотид, кодирующий полипептид, по любому из пп. 25-29, или комбинацию по любому из пп. 31-34.
36. Полинуклеотид по п. 35, отличающийся тем, что одна или несколько или все полипептид-кодирующие области полинуклеотида были кодон-модифицированы для экспрессии в растительной клетке, по сравнению с соответствующей полипептид-кодирующей областью кодирования природного полинуклеотида в бактерии.
37. Полинуклеотид по п. 35 или 36, дополнительно содержащий промотор, функционально связанный с полинуклеотидом, кодирующим каждый полипептид.
38. Полинуклеотид по любому из пп. 35-37, который присутствует в растительной клетке, дрожжевой клетке или бактериальной клетке.
39. Полинуклеотид по п. 38, который интегрирован в ядерный геном растительной клетки и/или экспрессирован в ядро растительной клетки.
40. Применение полинуклеотида по любому из пп. 35-39 для продуцирования трансгенной растительной клетки.
41. Растение для экспрессии полипептида NifD, отличающееся тем, что растение является трансгенным в отношении полинуклеотида по любому из пп. 35-39.
42. Часть растения для экспрессии полипептида NifD, отличающаяся тем, что часть растения является трансгенной в отношении полинуклеотида по любому из пп. 35-39.
43. Часть растения по п. 23 или 42, отличающаяся тем, что представляет собой семя.
44. Способ производства муки, муки крупного помола, крахмала, масла, кормовой муки из семян или другого продукта, полученного из семян, при этом способ включает: экстрагирование муки, муки крупного помола, крахмала, масла или другого продукта или получение кормовой муки из части растения по п. 43.
45. Пищевой продукт, полученный из трансгенного растения по пп. 21, 22 или 41, и/или части растения по пп. 23, 24, 42 или 43, где продукт содержит полипептид или MPP-расщепленный продукт, по любому из пп. 25-30 и/или полинуклеотид по любому из пп. 35-39.
46. Способ приготовления пищевого продукта, способ, включающий: смешивание части растения по п. 43 или муки, муки грубого помола, крахмала, масла или другого продукта из семян с другим пищевым ингредиентом.
47. Способ кормления животного, включающий предоставление животному растения или его части по пп. 21, 22 или 41, части растения по пп. 23, 24, 42 или 43 или продукта по п. 45.
| US 20180297905 A1, 18.10.2018 | |||
| WO 2018141030 A1, 09.08.2018 | |||
| RU 2017113250 A, 17.10.2018 | |||
| CN 0108291219 A, 17.07.2018. |
Авторы
Даты
2025-01-30—Публикация
2020-03-06—Подача