Изобретение относится к молекулярной биологии и касается набора олигонуклеотидов для определения микроРНК: hsa-miR-134-5р, hsa-miR-106b-5p, hsa-miR-191 и анализа относительной экспрессии данных микроРНК в плазме крови.
Данное изобретение предназначено для анализа экспрессии hsa-miR-134-5р, hsa-miR-106b-5p, hsa-miR-191 методом количественной ПЦР в два этапа. Первый этап заключается в проведении обратной транскрипции с матрицы микроРНК. Второй этап заключается в постановке и анализе количественной ПЦР в реальном времени с использованием технологии TaqMan. Экспрессионный анализ может быть использован в качестве метода для поиска биомаркеров заболеваний различной этиологии, в том числе для диагностики неврологических заболеваний, таких как эпилепсия.
В настоящее время анализ экспрессии микроРНК применяется в фундаментальных исследованиях эпигенетической регуляции экспрессии ряда таргетных генов, а также для поисковых исследований диагностических и прогностических биомаркеров ряда неврологических заболеваний.
Известна разработка набора праймеров и зондов для детекции микроРНК и последующего количественного анализа с использованием ПЦР в реальном времени, представляющая собой набор олигонуклеотидов, в числе которых праймер для обратной транскрипции микроРНК S-oligo(dT) [Патент на изобретение EP2700719A1 «Method and primers for detection of mirna, and application thereof», опубл. 26.02.2014]. В данном методе микроРНК сначала подвергается подиаденилированию для удлинения первоначальной последовательности с использованием фермента поли-А-полимеразы. После чего она обратно транскрибируется с использованием S-Oligo(dT)-праймера. Праймер S-Oligo(dT) для количественного анализа микроРНК состоит из четырех сегментов: в направлении от 5' к 3' концам, от 14 до 20 нуклеотидов последовательности для связывания универсального обратного праймера для ПЦР, от 14 до 20 нуклеотидов последовательности для связывания универсального зонда, от 8 до 30 нуклеотидов последовательности олиго(dT), за которыми следуют от 3 до 8 специфических нуклеотидов, которые комплементарны 3'-концу исследуемой микроРНК. Недостатком данной разработки является необходимости предварительного полиаденилирования микроРНК, что увеличивает время и стоимость реакции. Также к недостаткам можно отнести короткую последовательность специфических нуклеотидов (3-8). Наличие подобной структуры может привести к недостаточной специфичности обратной транскрипции, а также низкой температуре отжига праймера, и, соответственно необходимости проведения процедуры обратной транскрипции в два этапа.
Также известна разработка представляющая собой набор олигонуклеотидов для анализа экспрессии микроРНК, представляющая собой набор праймеров для обратной транскрипции, а также прямого и обратного праймера для ПЦР в реальном времени [Патент на изобретение US20160177376A1 «Detection method of micro-rna with high specificity», опубл. 23.06.2016]. В данном методе методе микроРНК сначала подвергается подиаденилированию для удлинения первоначальной последовательности с использованием фермента поли-А-полимеразы. Далее проводится процедура обратной транскрипции с использованием праймера, содержащего последовательность, комплементарную поли-А-хвосту. Последующий анализ проводится с использованием прямого праймера, комплементарного последовательности исследуемой микроРНК и обратного праймера. Анализ проводится с использованием интеркалирующего красителя в режиме плавления. Недостатком данной разработки является необходимости предварительного полиаденилирования микроРНК, что увеличивает время и стоимость реакции. Также к недостаткам можно отнести, отсутствие последовательности, комплементарной последовательности исследуемой микроРНК в составе праймера для обратной транскрипции, что может привести к неспецифичному отжиге в ходе процедуры обратной транскрипции.
Наиболее близким техническим решением, выбранным в качестве прототипа, является способ количественного определения микроРНК методом ПЦР-РВ с использованием стебель-петля (stem-loop)-праймеров [Chen C, Ridzon DA, Broomer AJ, Zhou Z, Lee DH, Nguyen JT, Barbisin M, Xu NL, Mahuvakar VR, Andersen MR, Lao KQ, Livak KJ, Guegler KJ. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Res. 2005 Nov 27;33(20):e179. doi: 10.1093/nar/gni178. PMID: 16314309; PMCID: PMC1292995.]. В данном методе используется праймер для обратной транскрипции типа стебель-петля, представляющий собой последовательность, комплементарную целевой микроРНК длиной 6 нуклеотидов и петлевой домен длиной до 44 нуклеотидов, формирующий шпилечную структуру ДНК, что удлиняет целевую микроРНК и делает возможным последующее проведение количественной ПЦР.
Недостатком данного метода является короткий специфический микроРНК фрагмент диной всего 6 нуклеотидов, что может привести к неспецифичности отжига праймера. Также температура отжига специфичного участка варьирует в диапазоне от 10 до 18 градусов Цельсия, что приводит к необходиомти двухэтапной обратной транскрипции: отжиг праймеров при 12-18 градусах и обратная транскрипция при 40 градусах. Так как оптимум работы фермента MMLV-ревертазы приходится на 40 градусов Цельсия.
Преимуществом нашего изобретения является увеличение комплементарного микроРНК фрагмента до 12 нуклеотидов (фрагменты, выделенные жирным шрифтом), что уменьшает вероятность неспецифического отжига праймеров. Также, преимуществом является повышение температуры отжига праймеров для обратной транскрипции до 38-40 градусов Цельсия, что позволяет провести отжиг праймеров и обратную транскрипцию в одну реакцию.
Технический результат изобретения заключается в повышении специфичности количественного анализа микроРНК, снижению временных затрат на проведение анализа. Таким образом, диагностика эпилепсии становится более точной и достоверной. При этом диагностика является дифференцированной и позволяет определить не только наличие эпилепсии, но и ее вид.
Технический результат изобретения достигается проведением процедуры обратной транскрипции с использованием входящих в набор stem-loop праймеров для получения комплементарных мирокРНК-134-5р, микроРНК-106b-5p, микроРНК-191 фрагментов ДНК. Новым является нуклеотидный состав синтетических олигонуклеотидов, входящих в состав stem-loop праймера, определяющий специфичность исследования таргетной микроРНК, а также сбалансированность по температурам отжига, находящихся в диапазоне 38-40 градусов Цельсия для проведения обратной транскрипции.
Анализ экспрессии проводится при помощи метода ПЦР в реальном времени с использованием разработанных прямого и обратного праймера, а также флуоресцентного зонда. Температуры отжига прямого и обратного праймеров для ПЦР находятся в диапазоне 58-60 градусов Цельсия, а также 65 градусов Цельсия для флуоресцентного зонда.
МикроРНК-191 является референсным геном, относительно которого анализируется экспрессия микроРНК 134-5р, микроРНК 106b-5р.
После окончания проведения ПЦР в реальном времени считываются пороговые циклы для каждой микроРНК (cycle threshold - ct). Расчеты относительной экспрессии таргетных микроРНК проводятся с использованием метода delta ct [Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 2001 Dec;25(4):402-8. doi: 10.1006/meth.2001.1262. PMID: 11846609.], в ходе которого из порогового цикла таргетной микроРНК (микроРНК134-5р и микроРНК106b-5p) вычитается пороговый цикл референсного гена (микроРНК-191).
Прописанные последовательности олигонуклеотидов получены путем анализа депонированных в базе данных MiRBase (https://mirbase.org) последовательностей микроРНК 134-5р, 106b-5р, и 191. Анализ полученных праймеров был проведен с использованием программного обеспечения PrimerSelect (https://bio.tools/primerselect). Температуры плавления были проанализированы с использованием программного обеспечения OligoCalc (http://biotools.nubic.northwestern.edu/OligoCalc.html).
Полученные наборы состоят из четырех синтетических олигонуклеотидов каждый:
Набор олигонуклеотидов для анализа экспрессии микроРНК-134-5р, состоящий из четырех синтетических последовательностей:
Праймер для обратной транскрипции типа стебель-петля (stem-loop):
CAGGCGATAGTCCGATGCATGCGCATGCATCGGACTATCGCCTGTT GGTGACTAGGTG
F-праймер для ПЦР:
TCCTGTGGGCCACCTAGTCAC
R-праймер для ПЦР:
CGTACGTAGCCTGATAGCGGAC
Зонд для ПЦР:
FAM CAACAGGCGATAGTCCGATGCATGC BHQ1.
2) Набор олигонуклеотидов для анализа экспрессии микроРНК-106b-5р, состоящий из четырех синтетических последовательностей:
Праймер для обратной транскрипции типа стебель-петля (stem-loop):
CAGGCGATAGTCCGATGCATGCGCATGCATCGGACTATCGCCTGAT CTGCACTGTCAG
F-праймер для ПЦР:
TGACAGTCGTGAAATAATGTATGTC
R-праймер для ПЦР:
CGTACGTAGCCTGATAGCGGAC
Зонд для ПЦР:
FAM CAGATGTCGTATCCAGTGCGAATAC BHQ1.
3) Набор олигонуклеотидов для анализа экспрессии микроРНК-191, состоящий из четырех синтетических последовательностей:
Праймер для обратной транскрипции типа стебель-петля (stem-loop):
CAGGCGATAGTCCGATGCATGCGCATGCATCGGACTATCGCCTG GGGGACGAAATC
F-праймер для ПЦР:
GCTTTAGGTTCGCGTCCTGT
R-праймер для ПЦР:
CGTACGTAGCCTGATAGCGGAC
Зонд для ПЦР:
FAM CCCCGTCGTATCCAGTGCGAATAC BHQ1.
Осуществление способа. Анализ экспрессии микроРНК проводится следующим образом. Кровь из кубитальной вены (1 вакутейнер 4 мл) исследуемого субъекта центрифугируется в два этапа по стандартному протоколу [Zheng XH, Cui C, Zhou XX, Zeng YX, Jia WH. Centrifugation: an important pre-analytic procedure that influences plasma microRNA quantification during blood processing. Chin J Cancer. 2013 Dec;32(12):667-72.]. Следующим этапом проводится выделение тотальной РНК плазмы с использованием стандартных протоколов для выделения РНК методом аффинной сорбции на силика-частицах [Salvo-Chirnside E, Kane S, Kerr LE. Protocol: high throughput silica-based purification of RNA from Arabidopsis seedlings in a 96-well format. Plant Methods. 2011 Dec 2;7(1):40. doi: 10.1186/1746-4811-7-40. PMID: 22136293; PMCID: PMC3305896].
Далее проводится процедура обратной транскрипции таргетных микроРНК, в ходе которой выделенная тотальная РНК плазмы инкубируется в буфере для обратной транскрипции, содержащем необходимую концентрацию ионов, смесь дезоксинуклеозидтрифосфатов, дитиотретил, с добавлением разработанного stem-loop праймера и MMLV-ревертазы в течение 40-60 минут при температуре 40 градусов Цельсия. После этапа синтеза комплементарной ДНК (кДНК), пробирки с полученным раствором прогреваются до 70 градусов Цельсия в течении 10 минут, для денатурации ревертазы.
Анализ экспрессии проводится методом ПЦР в реальном времени. Полученная кДНК добавляется в стандартный буфер для ПЦР в реальном времени, содержащий ионы магния, dNTPS, ddH2O, в который также раскапываются разработанные прямой и обратный праймеры, а также флуоресцентный зонд для ПЦР. ПЦР проводится в режиме: 90 градусов Цельсия - 3 минуты - предварительный прогрев, а далее 40 циклов: 95 градусов Цельсия - 15 секунд - денатураций, 60 градусов Цельсия - 60 секунд - отжиг и элонгация.
Анализ экспрессии проводится с использованием стандартного метода delta-delta ct. После проведения ПЦР в реальном времени мы получаем параметр ct (пороговый цикл) для каждой мироРНК. Для получения параметра delta ct из параметра ct исследуемой микроРНК вычитается параметр ct референсного гена. В качестве референсного гена выступает микроРНК-191, т.к. она является одной из наиболее стабильных плазменных микроРНК [Donati, S.; Ciuffi, S.; Brandi, M.L. Human Circulating miRNAs Real-time qRT-PCR-based Analysis: An Overview of Endogenous Reference Genes Used for Data Normalization. Int. J. Mol. Sci. 2019, 20, 4353].
Delta- ct данные по каждой микроРНК для каждого пациента и здорового волонтера сравниваются с использованием критерия Манна-Уитни для определения наличия либо отсутствия статистически значимых различий. Для расчета де Delta-delta- ct, высчитывается среднее Delta- ct для когорты пациентов и когорты контроля, из среднего Delta- ct когорты пациентов отнимается среднее дельта ct когорты контроля. Расчет параметра Fold (кратность изменения) проводится по формуле: 2^delta-delta ct.
Изобретение поясняется чертежами.
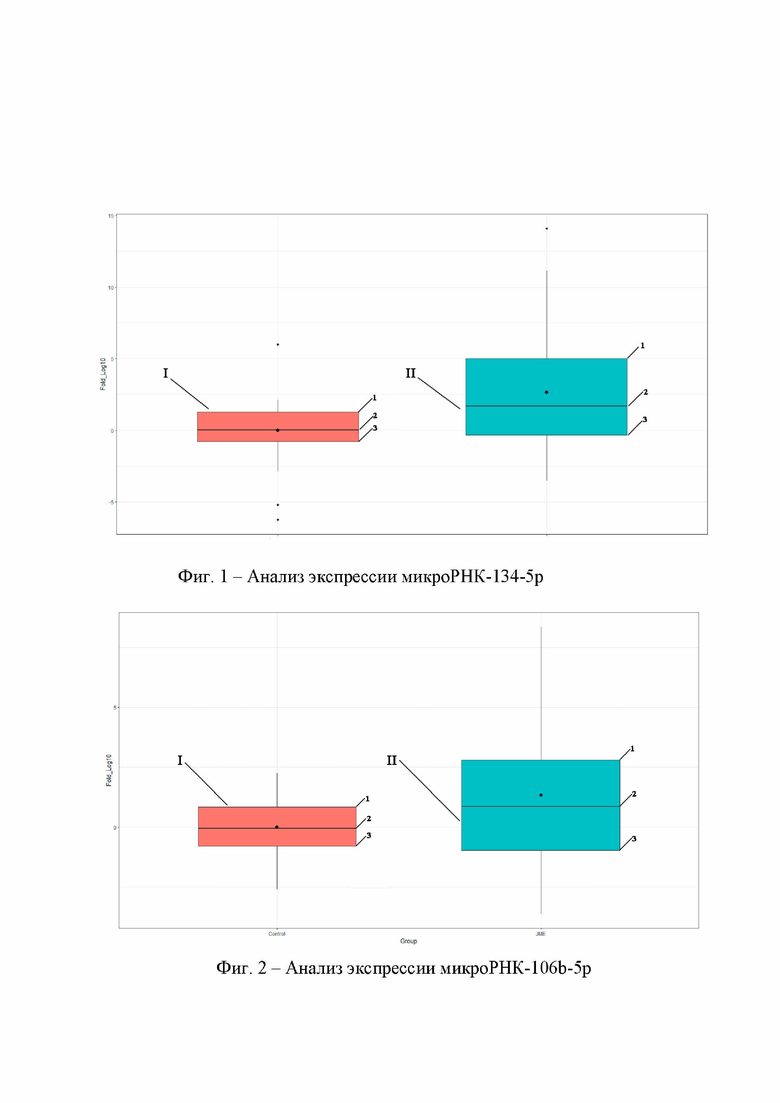
На фиг. 1 представлено сравнение экспрессии микроРНК hsa-miR-134-5p у пациентов с мезиальной височной эпилепсией и здоровых волонтеров, проведенное с использованием разработанного набора олигонуклеотидов.
Фиг. 1 демонстрирует дискриминацию группы пациентов с ВЭ от группы контроля по параметру delta-delta ct.
Для сравнения двух независимых выборок использовался непараметрический критерий Манна-Уитни, расчетный р-критерий равнялся 0.0006, что говорит о значимости различий между выборками. Так как кратность изменения Fold более 1, и в данном случае равна = 14.01, это свидетельствует о значительном повышении экспрессии микроРНК hsa-miR-134-5p у группы пациентов с мезиальной височной эпилепсией по сравнению с группой контроля.
На Фиг. 1 изображены результаты анализа экспрессии микроРНК-134-5р: экспрессия микроРНК в контрольной группе здоровых волонтеров (I), экспрессия микроРНК в группе пациентов с мезиальной височной эпилепсией(II). Условные обозначения: 1 - 75-й процентиль; 2 - Медианное значение; 3 - 25-й процентиль. Первый квартиль (25-й процентиль) определяется как среднее число между наименьшим числом (минимумом) и значением, находящимся между 25-м и 75-м процентилями выборки (которое отсекает первую четверть всех выборок). Его также называют нижним квартилем, поскольку 25% данных находятся ниже этой точки.
Второй квартиль (медианное значение) - это медиана набора данных; таким образом, 50% данных лежат ниже этой точки.
Третий квартиль (75-й процентиль) - это среднее значение между значением, отсекающим последнюю четверть выборок, и самым высоким значением (максимумом) набора данных. Он известен как верхний квартиль, поскольку 75% данных находятся ниже этой точки. Таким образом, на графике отображен массив значений экспрессии, полученной для каждой из двух исследуемых групп. Расстояния между различными частями ящика позволяют определить степень разброса данных в исследуемых группах.
В группе контроля (I) получены следующие численные значения: 1=1.3; 2=0.1; 3=-0.8.
В группе пациентов с мВЭ (II) получены следующие численные значения: 1=5.2; 2=1.7; 3=-0.4.
Полученные в ходе исследования данные свидетельствуют о возможности использования микроРНК hsa-miR-134-5p в качестве биомаркера мезиальной височной эпилепсии, что позволяет его использование в качестве средства дополнительной диагностики.
Расчеты проводились с использованием Python numpy, seaborn, matplotlib.
На фиг. 2 представлено сравнение экспрессии микроРНК hsa-miR-106b-5p у пациентов с юношеской миоклонической эпилепсией и здоровых волонтеров, проведенное с использованием разработанного набора олигонуклеотидов, Фиг. 1 демонстрирует дискриминацию группы пациентов с ВЭ от группы контроля по параметру delta-delta ct.
Для сравнения двух независимых выборок использовался непараметрический критерий Манна-Уитни, расчетный р-критерий равнялся 0.025, что говорит о значимости различий между выборками. Fold = 3.03, что свидетельствует о значительном повышении экспрессии микроРНК hsa-miR-106b-5p у группы пациентов с мезиальной височной эпилепсией по сравнению с группой контроля. На Фиг. 1 изображены результаты анализа экспрессии микроРНК-106b-5p: экспрессия микроРНК в контрольной группе здоровых волонтеров (I), экспрессия микроРНК в группе пациентов с юношеской миоклонической эпилепсией (II). Условные обозначения: 1 - 75-й процентиль; 2 - Среднее значение; 3 - 25-й процентиль.
В группе контроля (I) получены следующие численные значения: 1=0.8; 2=-0.1; 3=-0.8.
В группе пациентов с ЮМЭ (II) получены следующие численные значения: 1=2.8; 2=0.9; 3=-1.
Полученные в ходе исследования данные свидетельствуют о возможности использования микроРНК hsa-miR-106b-5p в качестве биомаркера юношеской миоклонической эпилепсии, что позволяет его использование в качестве средства дополнительной диагностики.
Расчеты проводились с использованием Python numpy, seaborn, matplotlib.
Пример 1. Исследовалась когорта пациентов с мезиальной височной эпилепсией (ВЭ), состоящая из 62 человек, со средним возрастом 36 лет (75%: 42.5; 50%: 36; 25%: 28.5), с 51.6% - женщин и 49.6% - мужчин, а также когорта здоровых волонтеров (Контроль), состоящая из 50 человек, со средним возрастом 31.06 лет (75%: 34.75; 50%: 27; 25%: 26), с 76% - женщин и 24 % - мужчин. Кровь из кубитальной вены (1 вакутейнер 4 мл) исследуемого субъекта центрифугируется в два этапа по стандартному протоколу. Следующим этапом проводится выделение тотальной РНК плазмы с использованием стандартных протоколов для выделения РНК методом аффинной сорбции на силика-частицах. Далее проводится процедура обратной транскрипции таргетных микроРНК, в ходе которой выделенная тотальная РНК плазмы инкубируется в буфере для обратной транскрипции, содержащего необходимую концентрацию ионов, смесь дезоксинуклеозидтрифосфатов, дитиотретил, с добавлением разработанного stem-loop праймера для hsa-miR-134-5p, а также has-miR-191, использованный в качестве референсного гена и MMLV-ревертазы в течении 40-60 минут при температуре 40 градусов Цельсия. После этапа синтеза комплементарной ДНК (кДНК), пробирки с полученным раствором прогреваются до 70 градусов Цельсия в течении 10 минут, для денатурации ревертазы.
Далее проводится ПЦР в реальном времени, для чего раствор, содержащий кДНК, раскапывается в стандартный буфер для ПЦР в реальном времени, содержащий Taq-полимеразу и ионы магния. В ПЦР-микс добавляются разработанные прямой и обратный праймеры, а также флуоресцентный зонд для ПЦР. ПЦР проводится в режиме: 90 градусов Цельсия - 3 минуты - предварительный прогрев, а далее 40 циклов: 95 градусов Цельсия - 15 секунд - денатурация, 60 градусов Цельсия - 60 секунд - отжиг и элонгация. Анализ экспрессии проводится методом delta-delta ct. Полученные результаты позволяют дискриминировать пациентов с височной эпилепсией от здорового контроля (Фиг. 1).
Анализ экспрессии проводится с использованием стандартного метода delta-delta ct. Delta- ct данные по микроРНК-134-5р и микроРНК-191 для каждого пациента и здорового волонтера сравниваются с использованием критерия Манна-Уитни для определения наличия либо отсутствия статистически значимых различий. Для расчета де Delta-delta- ct, высчитывается среднее Delta- ct для когорты пациентов и когорты контроля, из среднего Delta- ct когорты пациентов отнимается среднее дельта ct когорты контроля. Расчет параметра Fold (кратность изменения) проводится по формуле: 2^delta-delta ct. И составляет 14.01.
Пример 2. Исследовалась когорта пациентов с юношеской миоклонической эпилепсией (ЮМЭ), состоящая из 47человек, со средним возрастом 28.04 (75%: 33; 50%: 26; 25%:20), с 68.08% - женщин и 31.9 % - мужчин, а также когорта здоровых волонтеров (Контроль), а также когорта здоровых волонтеров (Контроль), состоящая из 50 человек, со средним возрастом 31.06 лет (75%: 34.75; 50%: 27; 25%: 26), с 76% - женщин и 24 % - мужчин. Кровь из кубитальной вены исследуемого субъекта центрифугируется в два этапа Кровь из кубитальной вены (1 вакутейнер 4 мл) исследуемого субъекта центрифугируется в два этапа по стандартному протоколу. Следующий этапом проводится выделение тотальной РНК плазмы с использованием стандартных протоколов для выделения РНК методом аффинной сорбции на силика-частицах. Далее проводится процедура обратной транскрипции таргетных микроРНК, в ходе которой выделенная тотальная РНК плазмы инкубируется в буфере для обратной транскрипции, содержащего необходимую концентрацию ионов, смесь дезоксинуклеозидтрифосфатов, дитиотретил, с добавлением разработанного stem-loop праймера для hsa-miR-106b-5p, а также has-miR-191, использованный в качестве референсного гена и MMLV-ревертазы в течении 40-60 минут при температуре 40 градусов Цельсия.
Далее проводится ПЦР в реальном времени для чего раствор, содержащий к ДНК раскапывается в стандартный буфер для ПЦР в реальном времени, содержащий Taq-полимеразу и ионы магния. В ПЦР-микс добавляются разработанные прямой и обратный праймеры, а также флуоресцентный зонд для ПЦР. ПЦР проводится в режиме: 90 градусов Цельсия - 3 минуты - предварительный прогрев, а далее 40 циклов: 95 градусов Цельсия - 15 секунд - денатурация, 60 градусов Цельсия - 60 секунд - отжиг и элонгация. Анализ экспрессии проводится методом delta-delta ct. Полученные результаты позволяют дискриминировать пациентов с височной эпилепсией от здорового контроля (Фиг. 1).
Анализ экспрессии проводится с использованием стандартного метода delta-delta ct. Delta- ct данные по микроРНК-134-5р и микроРНК-191 для каждого пациента и здорового волонтера сравниваются с использованием критерия Манна-Уитни для определения наличия либо отсутствия статистически значимых различий. Для расчета де Delta-delta- ct, высчитывается среднее Delta- ct для когорты пациентов и когорты контроля, из среднего Delta- ct когорты пациентов отнимается среднее дельта ct когорты контроля. Расчет параметра Fold (кратность изменения) проводится по формуле: 2^delta-delta ct. И составляет: 3.803.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="ru"
nonEnglishFreeTextLanguageCode="ru" dtdVersion="V1_3"
fileName="Олигонуклеотиды для диагностики эпилепсии методом
количественной ПЦР.xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2023-12-19">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText></ApplicationNumberText>
<FilingDate>2023-12-19</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>1</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное образовательное учреждение высшего образования
"Красноярский государственный медицинский университет им. проф.
В.Ф. Войно-Ясенецкого" Министерства здравоохранения Российской
Федерации</ApplicantName>
<ApplicantNameLatin>Federal State Budgetary Educational Institution
of Higher Education "Prof. V.F. Voino-Yasenetsky Krasnoyarsk
State Medical University" of Healthcare of the Russian
Federation</ApplicantNameLatin>
<InventorName languageCode="ru">Тимечко Елена
Евгеньевна</InventorName>
<InventorNameLatin>Timechko Elena Evgenyevna</InventorNameLatin>
<InventionTitle languageCode="ru">Олигонуклеотиды для диагностики
эпилепсии методом количественной ПЦР</InventionTitle>
<SequenceTotalQuantity>12</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caggcgatagtccgatgcatgcgcatgcatcggactatcgcctgttggt
gactaggtg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcctgtgggccacctagtcac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtacgtagcctgatagcggac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q9">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>6-Carboxyfluorescein</INSDQualifier_value>
<NonEnglishQualifier_value>6-Карбоксифлуоресцеин</NonEnglishQua
lifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Black Hole Quencher
1</INSDQualifier_value>
<NonEnglishQualifier_value>Black Hole Quencher
1</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caacaggcgatagtccgatgcatgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caggcgatagtccgatgcatgcgcatgcatcggactatcgcctgatctg
cactgtcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgacagtcgtgaaataatgtatgtc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtacgtagcctgatagcggac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q19">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>6-Carboxyfluorescein</INSDQualifier_value>
<NonEnglishQualifier_value>6-Карбоксифлуоресцеин</NonEnglishQua
lifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Black Hole Quencher
1</INSDQualifier_value>
<NonEnglishQualifier_value>Black Hole Quencher
1</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cagatgtcgtatccagtgcgaatac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>56</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..56</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caggcgatagtccgatgcatgcgcatgcatcggactatcgcctggggga
cgaaatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctttaggttcgcgtcctgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtacgtagcctgatagcggac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q29">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>6-Carboxyfluorescein</INSDQualifier_value>
<NonEnglishQualifier_value>6-Карбоксифлуоресцеин</NonEnglishQua
lifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Black Hole Quencher
1</INSDQualifier_value>
<NonEnglishQualifier_value>Black Hole Quencher
1</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccccgtcgtatccagtgcgaatac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Набор реагентов для определения малых рибонуклеиновых кислот (микроРНК) при диагностике множественной миеломы | 2024 |
|
RU2836054C1 |
| Набор олигодезоксирибонуклеотидных праймеров для диагностики сердечно-сосудистых заболеваний и выявления риска их развития на основе биомолекулярного маркера микроРНК hsa-miR-143-5p методом количественной ПЦР и его применение | 2025 |
|
RU2838258C1 |
| Рекомбинантная плазмидная ДНК, кодирующая микроРНК hsa-miR-145-5p, и ее применение в качестве положительного контроля при ПЦР-РВ для диагностики сердечно-сосудистых заболеваний и выявления риска их развития | 2025 |
|
RU2837874C1 |
| Рекомбинантная плазмидная ДНК, кодирующая микроРНК hsa-miR-143-5p, и ее применение в качестве положительного контроля при ПЦР-РВ для диагностики сердечно-сосудистых заболеваний и выявления риска их развития | 2025 |
|
RU2837872C1 |
| Рекомбинантная плазмидная ДНК, кодирующая микроРНК hsa-mir-143-3p, и ее применение в качестве положительного контроля при ПЦР-РВ для диагностики сердечно-сосудистых заболеваний и выявления риска их развития | 2025 |
|
RU2837882C1 |
| Рекомбинантная плазмидная ДНК, кодирующая микроРНК hsa-miR-145-3p, и ее применение в качестве положительного контроля при ПЦР-РВ для диагностики сердечно-сосудистых заболеваний и выявления риска их развития | 2025 |
|
RU2837873C1 |
| Набор олигодезоксирибонуклеотидных праймеров для диагностики сердечно-сосудистых заболеваний и выявления риска их развития на основе биомолекулярного маркера микроРНК hsa-miR-143-3p методом количественной ПЦР и его применение | 2025 |
|
RU2838257C1 |
| Способ прогнозирования привычной невынашиваемости беременности по экспрессии генов микроРНК в биопсийном материале эндометрия | 2025 |
|
RU2837879C1 |
| Набор олигодезоксирибонуклеотидных праймеров для диагностики сердечно-сосудистых заболеваний и выявления риска их развития на основе биомолекулярного маркера микроРНК hsa-miR-145-3p методом количественной ПЦР и его применение | 2025 |
|
RU2838260C1 |
| Способ диагностики глиальных опухолей головного мозга высокой степени злокачественности | 2020 |
|
RU2742413C1 |
Изобретение относится к биотехнологии. Раскрыт набор олигонуклеотидов для определения микроРНК hsa-miR-134-5р, hsa-miR-106b-5p, hsa-miR-191 и анализа относительной экспрессии данных микроРНК в плазме крови. Набор имеет следующий состав: олигонуклеотиды для анализа экспрессии микроРНК-134-5р, олигонуклеотиды для анализа экспрессии микроРНК-106b-5р и олигонуклеотиды для анализа экспрессии микроРНК-191. При этом для анализа экспрессии микроРНК-134-5р используют: праймер для обратной транскрипции CAGGCGATAGTCCGATGCATGCGCATGCATCGGACTATCGCCTGTTGGTGACTAGGT, прямой F-праймер TCCTGTGGGCCACCTAGTCAC, R-праймер CGTACGTAGCCTGATAGCGGAC, зонд FAM CAACAGGCGATAGTCCGATGCATGC BHQ1. Для анализа экспрессии микроРНК-106b-5р используют: праймер для обратной транскрипции CAGGCGATAGTCCGATGCATGCGCATGCATCGGACTATCGCCTGATCTGCACTGTCA, F-праймер TGACAGTCGTGAAATAATGTATGTC, R-праймер CGTACGTAGCCTGATAGCGGAC, зонд FAM CAGAT GTCGTATCCAGTGCGAATAC BHQ1. Для анализа экспрессии микроРНК-191 используют: праймер для обратной транскрипции CAGGCGATAGTCCGATGCATGCGCATGCATCGGACTATCGCCTGGGGGACGAAATC, F-праймер GCTTTAGGTTCGCGTCCTGT, R-праймер CGTACGTAGCCTGATAGCGGAC, зонд FAM CCCCGTCGTATCCAGTGCGAATAC BHQ1. МикроРНК-191 является референсным геном, относительно которого анализируется экспрессия микроРНК 134-5р, микроРНК 106b-5р. Технический результат изобретения заключается в повышении специфичности количественного анализа микроРНК, снижении временных затрат на проведение анализа. При этом диагностика является дифференцированной и позволяет определить не только наличие эпилепсии, но и ее вид. Так, микроРНК hsa-miR-106b-5p используется в качестве биомаркера юношеской миоклонической эпилепсии, а микроРНК hsa-miR-134-5p - в качестве биомаркера мезиальной височной эпилепсии. 2 ил., 2 пр.
Набор олигонуклеотидов для анализа экспрессии микроРНК-134-5р, микроРНК-106b-5p и микроРНК-191 методом количественной полимеразной цепной реакции, проводимой в режиме реального времени, имеющий следующий нуклеотидный состав:
1) набор олигонуклеотидов для анализа экспрессии микроРНК-134-5р, состоящий из четырех синтетических последовательностей:
- праймер для обратной транскрипции
CAGGCGATAGTCCGATGCATGCGCATGCATCGGACTATCGCCTGTTGGTGACTAGGTG;
- прямой F-праймер для ПЦР TCCTGTGGGCCACCTAGTCAC;
- R-праймер для ПЦР CGTACGTAGCCTGATAGCGGAC;
- зонд для ПЦР FAM CAACAGGCGATAGTCCGATGCATGC BHQ1;
2) набор олигонуклеотидов для анализа экспрессии микроРНК-106b-5р, состоящий из четырех синтетических последовательностей:
- праймер для обратной транскрипции
CAGGCGATAGTCCGATGCATGCGCATGCATCGGACTATCGCCTGATCTGCACTGTCAG;
- F-праймер для ПЦР TGACAGTCGTGAAATAATGTATGTC;
- R-праймер для ПЦР CGTACGTAGCCTGATAGCGGAC;
- зонд для ПЦР FAM CAGAT GTCGTATCCAGTGCGAATAC BHQ1;
3) набор олигонуклеотидов для анализа экспрессии микроРНК-191, состоящий из четырех синтетических последовательностей:
- праймер для обратной транскрипции
CAGGCGATAGTCCGATGCATGCGCATGCATCGGACTATCGCCTGGGGGACGAAATC;
- F-праймер для ПЦР GCTTTAGGTTCGCGTCCTGT;
- R-праймер для ПЦР CGTACGTAGCCTGATAGCGGAC;
- зонд для ПЦР FAM CCCCGTCGTATCCAGTGCGAATAC BHQ1.
| Chen C., Ridzon D.A., Broomer A.J., Zhou Z., Lee D.H., Nguyen J.T., Barbisin M., Xu N.L., Mahuvakar V.R., Andersen M.R., Lao K.Q., Livak K.J., Guegler K.J | |||
| Real-time quantification of microRNAs by stem-loop RT-PCR | |||
| Nucleic Acids Res | |||
| Способ обработки целлюлозных материалов, с целью тонкого измельчения или переведения в коллоидальный раствор | 1923 |
|
SU2005A1 |
| US 20160177376 A1, 23.06.2016 | |||
| GB 201223244 D0, 06.02.2013 | |||
| НОСИЛКИ СПАСАТЕЛЬНЫЕ БЕСКАРКАСНЫЕ | 2018 |
|
RU2700719C1 |
Авторы
Даты
2024-03-11—Публикация
2023-12-19—Подача