ОБЛАСТЬ ТЕХНИКИ, К КОТОРОЙ ОТНОСИТСЯ ИЗОБРЕТЕНИЕ
Настоящее изобретение относится к новым структурам двухцепочечной РНК (дцРНК) и их применению в сайленсинге генов.
УРОВЕНЬ ТЕХНИКИ
Обычные молекулы дцРНК, применяемые для индукции РНК-интерференции (РНКи) в клетках животных, образуются при отжиге одноцепочечных смысловых и антисмысловых РНК или из самокомплементарных РНК, имеющих области комплементарности, которые позволяют образовываться структурам стебель-петли, также называемых шпилечной РНК (шРНК). В растениях трансгены, экспрессирующие длинные шпилечные РНК, как оказалось, обладали высокой эффективностью при стимуляции РНКи. Некоторые исследования длинной шРНК показали, что продукты РНК процессинга дцРНК, называемые малыми интерферирующими РНК (миРНК), которые образуются из последовательностей, расположенных ближе к концу петли шРНК, отличаются большей представленностью по сравнению с открытым концом структуры шРНК, что означает, что процессинг Dicer дцРНК стебля шРНК, по-видимому, начинается с конца петли. Вся петля может быть точно вырезана из структуры шРНК, что может давать дцРНК конец, с которым может связываться фермент РНКаза Dicer и начинать процессинг миРНК.
Несмотря на то, что индуцированный дцРНК сайленсинг генов оказался ценным инструментом для изменения фенотипа организма, существует потребность в альтернативных, предпочтительно улучшенных, молекулах дцРНК, которые можно применять для РНКи.
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
Авторы изобретения разработали новую конструкцию РНКи-индуцирующей молекулы, называемую в настоящем документе дцРНК с концевой петлей (кпРНК), которая имеет одну или более следующих особенностей: легко синтезируется, легче образует структуру дцРНК и индуцирует эффективный сайленсинг генов-мишеней в эукариотических клетках. Также кпРНК эффективна при нанесении на поверхность листьев растений.
В первом аспекте настоящего изобретения предложена молекула рибонуклеиновой кислоты (РНК), включающая первый РНК-компонент, второй РНК-компонент, который ковалентно связан с первым РНК-компонентом и, необязательно, одно или более, или все из: (i) соединительной рибонуклеотидной последовательности, которая ковалентно связывает первый и второй РНК-компоненты, (ii) 5'-лидерной последовательности и (iii) 3'-трейлерной последовательности,
где первый РНК-компонент состоит, в порядке от 5' к 3', из первого 5'-рибонуклеотида, первой последовательности РНК и первого 3'-рибонуклеотида, где первые 5' и 3' рибонуклеотиды спариваются друг с другом в молекуле РНК, где первая последовательность РНК включает первую смысловую рибонуклеотидную последовательность по меньшей мере из 20 последовательных рибонуклеотидов, первую последовательность петли по меньшей мере из 4 рибонуклеотидов и первую антисмысловую рибонуклеотидную последовательность по меньшей мере из 20 последовательных рибонуклеотидов, где первая антисмысловая рибонуклеотидная последовательность гибридизуется с первой смысловой рибонуклеотидной последовательностью в молекуле РНК, где первая антисмысловая рибонуклеотидная последовательность способна гибридизоваться с первой областью молекулы РНК-мишени,
где второй РНК-компонент ковалентно связан через соединительную рибонуклеотидную последовательность, если таковая присутствует, или напрямую, если соединительная рибонуклеотидная последовательность не присутствует, с первым 5'-рибонуклеотидом или первым 3'-рибонуклеотидом,
где второй РНК-компонент состоит, в порядке от 5' к 3', из второго 5'-рибонуклеотида, второй последовательности РНК и второго 3'-рибонуклеотида, где вторые 5' и 3' рибонуклеотиды спариваются друг с другом в молекуле РНК, где вторая последовательность РНК включает вторую смысловую рибонуклеотидную последовательность, вторую последовательность петли по меньшей мере из 4 рибонуклеотидов и вторую антисмысловую рибонуклеотидную последовательность, где вторая смысловая рибонуклеотидная последовательность гибридизуется со второй антисмысловой рибонуклеотидной последовательностью в молекуле РНК,
где 5'-лидерная последовательность, если таковая присутствует, состоит из рибонуклеотидной последовательности, которая ковалентно связана с первым 5'-рибонуклеотидом, если второй РНК-компонент связан с первым 3'-рибонуклеотидом, или со вторым 5'-рибонуклеотидом, если второй РНК-компонент связан с первым 5'-рибонуклеотидом, и
где 3'-трейлерная последовательность, если таковая присутствует, состоит из рибонуклеотидной последовательности, которая ковалентно связана со вторым 3'-рибонуклеотидом, если второй РНК-компонент связан с первым 3'-рибонуклеотидом, или с первым 3'-рибонуклеотидом, если второй РНК-компонент связан с первым 5'-рибонуклеотидом.
Во втором аспекте настоящего изобретения предложена молекула РНК, включающая первый РНК-компонент, второй РНК-компонент, который ковалентно связан с первым РНК-компонентом, и, необязательно, один или более, или все из: (i) соединительной рибонуклеотидной последовательности, которая ковалентно связывает первый и второй РНК-компоненты, (ii) 5'-лидерной последовательности и (iii) 3'-трейлерной последовательности,
где первый РНК-компонент состоит, в порядке от 5' к 3', из первого 5'-рибонуклеотида, первой последовательности РНК и первого 3'-рибонуклеотида, где первые 5' и 3' рибонуклеотиды спариваются, где первая последовательность РНК включает первую смысловую рибонуклеотидную последовательность, первую последовательность петли по меньшей мере из 4 рибонуклеотидов и первую антисмысловую рибонуклеотидную последовательность, где первая смысловая рибонуклеотидная последовательность и первая антисмысловая рибонуклеотидная последовательность состоят по меньшей мере из 20 последовательных рибонуклеотидов, при этом по меньшей мере 20 последовательных рибонуклеотидов первой смысловой рибонуклеотидной последовательности полностью спариваются по меньшей мере с 20 последовательными рибонуклеотидами первой антисмысловой рибонуклеотидной последовательности, где по меньшей мере 20 последовательных рибонуклеотидов первой смысловой рибонуклеотидной последовательности или по меньшей мере 20 последовательных рибонуклеотидов первой антисмысловой рибонуклеотидной последовательности идентичны по последовательности первой области молекулы РНК-мишени или ее комплементу, соответственно, или обоим,
где второй РНК-компонент ковалентно связан, через соединительную рибонуклеотидную последовательность, если таковая присутствует, с первым 5'-рибонуклеотидом или первым 3'-рибонуклеотидом,
где второй РНК-компонент состоит, в порядке от 5' к 3', из второго 5'-рибонуклеотида, второй последовательности РНК и второго 3'-рибонуклеотида, где вторые 5' и 3' рибонуклеотиды спариваются, где вторая последовательность РНК включает вторую смысловую рибонуклеотидную последовательность, вторую последовательность петли по меньшей мере из 4 рибонуклеотидов и вторую антисмысловую рибонуклеотидную последовательность, где вторая смысловая рибонуклеотидная последовательность спаривается со второй антисмысловой рибонуклеотидной последовательностью,
где 5'-лидерная последовательность, если таковая присутствует, состоит из последовательности рибонуклеотидов, которая ковалентно связана с первым 5'-рибонуклеотидом, если второй РНК-компонент связан с первым 3'-рибонуклеотидом, или со вторым 5'-рибонуклеотидом, если второй РНК-компонент связан с первым 5'-рибонуклеотидом, и
где 3'-трейлерная последовательность, если таковая присутствует, состоит из последовательности рибонуклеотидов, которая ковалентно связана со вторым 3'-рибонуклеотидом, если второй РНК-компонент связан с первым 3'-рибонуклеотидом, или с первым 3'-рибонуклеотидом, если второй РНК-компонент связан с первым 5'-рибонуклеотидом.
В предпочтительном варианте осуществления молекула РНК согласно изобретению является химерной молекулой РНК.
В третьем аспекте настоящего изобретения предложена химерная молекула рибонуклеиновой кислоты (РНК), включающая область двухцепочечной РНК (дцРНК), включающую смысловую рибонуклеотидную последовательность и антисмысловую рибонуклеотидную последовательность, которые способны гибридизоваться друг с другом с образованием области дцРНК, где:
i) смысловая рибонуклеотидная последовательность состоит из ковалентно связанных, в порядке от 5' к 3', первого 5'-рибонуклеотида, первой последовательности РНК и первого 3'-рибонуклеотида,
ii) антисмысловая рибонуклеотидная последовательность состоит из ковалентно связанных, в порядке от 5' к 3', второго 5'-рибонуклеотида, второй последовательности РНК и второго 3'-рибонуклеотида,
iii) первый 5'-рибонуклеотид спаривается со вторым 3'-рибонуклеотидом с образованием концевой пары оснований области дцРНК,
iv) второй 5'-рибонуклеотид спаривается с первым 3'-рибонуклеотидом с образованием концевой пары оснований области дцРНК,
v) от приблизительно 5% до приблизительно 40% рибонуклеотидов смысловой рибонуклеотидной последовательности и антисмысловой рибонуклеотидной последовательности, в общей сложности, либо спарены с образованием неканонических пар оснований, либо не спарены,
vi) область дцРНК не содержит 20 последовательных канонических пар оснований,
vii) молекула РНК способна к процессингу в эукариотической клетке или in vitro, при этом антисмысловая рибонуклеотидная последовательность расщепляется с получением коротких молекул антисмысловой РНК (асРНК) длиной 20-24 рибонуклеотидов,
viii) молекула РНК или по меньшей мере некоторые молекулы асРНК, или и те, и другие, способны к снижению экспрессии или активности молекулы РНК-мишени в эукариотической клетке, и
ix) молекула РНК может быть получена ферментативно при транскрипции in vitro и/или в клетке.
В четвертом аспекте настоящего изобретения предложена химерная молекула РНК, включающая первый РНК-компонент и второй РНК-компонент, который ковалентно связан с первым РНК-компонентом,
где первый РНК-компонент включает первую область двухцепочечной РНК (дцРНК), включающую первую смысловую рибонуклеотидную последовательность и первую антисмысловую рибонуклеотидную последовательность, которые способны к гибридизации друг с другом с образованием первой области дцРНК, и первую промежуточную рибонуклеотидную последовательность по меньшей мере из 4 нуклеотидов, которая ковалентно связывает первую смысловую рибонуклеотидную последовательность и первую антисмысловую рибонуклеотидную последовательность,
где второй РНК-компонент включает вторую смысловую рибонуклеотидную последовательность, вторую антисмысловую рибонуклеотидную последовательность и вторую промежуточную рибонуклеотидную последовательность по меньшей мере из 4 рибонуклеотидов, которая ковалентно связывает вторую смысловую рибонуклеотидную последовательность и вторую антисмысловую рибонуклеотидную последовательность, где вторая смысловая рибонуклеотидная последовательность гибридизуется со второй антисмысловой рибонуклеотидной последовательностью в молекуле РНК,
где в первом РНК-компоненте:
i) первая смысловая рибонуклеотидная последовательность состоит по меньшей мере из 20 последовательных рибонуклеотидов, ковалентно связанных, в порядке от 5' к 3', первого 5'-рибонуклеотида, первой последовательности РНК и первого 3'-рибонуклеотида,
ii) первая антисмысловая рибонуклеотидная последовательность состоит по меньшей мере из 20 последовательных рибонуклеотидов, ковалентно связанных, в порядке от 5' к 3', второго 5'-рибонуклеотида, второй последовательности РНК и второго 3'-рибонуклеотида,
iii) первый 5'-рибонуклеотид спаривается со вторым 3'-рибонуклеотидом,
iv) второй 5'-рибонуклеотид спаривается с первым 3'-рибонуклеотидом,
v) от 5% до 40% рибонуклеотидов первой смысловой рибонуклеотидной последовательности и первой антисмысловой рибонуклеотидной последовательности, в общей сложности, либо спариваются с образованием неканонических пар оснований, либо не спариваются, и
vi) первая область дцРНК не содержит 20 последовательных канонических пар оснований,
где химерная молекула РНК способна к процессингу в эукариотической клетке или in vitro, при этом первая антисмысловая рибонуклеотидная последовательность расщепляется с получением короткой молекулы антисмысловой РНК (асРНК) длиной 20-24 рибонуклеотидов, и где:
(a) химерная молекула РНК или по меньшей мере некоторые молекулы асРНК, или и те, и другие, способны к снижению экспресссии или активности молекулы РНК-мишени в эукариотической клетке, или
(b) первая антисмысловая рибонуклеотидная последовательность включает последовательность по меньшей мере из 20 последовательных рибонуклеотидов, которая по меньшей мере на 50% идентична по последовательности области комплемента молекулы РНК-мишени, или
(c) и (a), и (b).
В первом, втором и четвертом аспектах первый 5'-рибонуклеотид и первый 3'-рибонуклеотид первого РНК-компонента спариваются с образованием пары оснований. Такая пара оснований определена в настоящем документе как концевая пара оснований области дцРНК, образованная при самогибридизации первого РНК-компонента. В варианте осуществления, где первая смысловая рибонуклеотидная последовательность ковалентно связана с первым 5'-рибонуклеотидом без каких-либо промежуточных нуклеотидов, и первая антисмысловая рибонуклеотидная последовательность ковалентно связана с первым 3'-рибонуклеотидом без каких-либо промежуточных нуклеотидов, первый 5'-рибонуклеотид непосредственно связан с одной из смысловой последовательности и антисмысловой последовательности, и первый 3'-рибонуклеотид непосредственно связан с другой смысловой последовательностью и антисмысловой последовательностью.
В предпочтительном варианте осуществления по меньшей мере 20 последовательных рибонуклеотидов первой антисмысловой рибонуклеотидной последовательности способны спариваться с нуклеотидами первой области молекулы РНК-мишени. В варианте осуществления первая смысловая рибонуклеотидная последовательность ковалентно связана с первым 5'-рибонуклеотидом без каких-либо промежуточных нуклеотидов, и/или первая антисмысловая рибонуклеотидная последовательность ковалентно связана с первым 3'-рибонуклеотидом без каких-либо промежуточных нуклеотидов.
В варианте осуществления молекула РНК включает соединительную рибонуклеотидную последовательность, где соединительная рибонуклеотидная последовательность является родственной по последовательности с геном-мишенью, или идентична, по меньшей мере частично, области молекулы РНК-мишени или ее комплементу. В предпочтительном варианте осуществления соединительная рибонуклеотидная последовательность вместе со смысловыми последовательностями в первом и втором РНК-компонентах являются частью одной непрерывной смысловой последовательности, или вместе с антисмысловыми последовательностями в первом и втором РНК-компонентах являются частью одной непрерывной антисмысловой последовательности. В варианте осуществления молекула РНК включает соединительную рибонуклеотидную последовательность, где соединительная рибонуклеотидная последовательность содержит меньше 20 рибонуклеотидов. В варианте осуществления соединительная рибонуклеотидная последовательность гибридизуется с молекулой РНК-мишени. В варианте осуществления соединительная рибонуклеотидная последовательность идентична части комплемента молекулы РНК-мишени. В варианте осуществления соединительная рибонуклеотидная последовательность содержит от 1 до 10 рибонуклеотидов.
В вариантах осуществления первого, второго или четвертого аспектов молекула РНК включает одно или более, или все из: (i) соединительной рибонуклеотидной последовательности, ковалентно связывающей первый и второй РНК-компоненты, (ii) 5'-удлиняющей последовательности и (iii), 3'-удлиняющей последовательности, где 5'-удлиняющая последовательность, если таковая присутствует, состоит из последовательности рибонуклеотидов, которая ковалентно связана с первым РНК-компонентом или со вторым РНК-компонентом, и где 3'-удлиняющая последовательность, если таковая присутствует, состоит из последовательности рибонуклеотидов, которая ковалентно связана со вторым РНК-компонентом или с первым РНК-компонентом, соответственно. В варианте осуществления первый РНК-компонент и второй РНК-компонент ковалентно связаны через соединительную рибонуклеотидную последовательность. В альтернативном варианте осуществления первый РНК-компонент и второй РНК-компонент непосредственно связаны без присутствия какой-либо соединительной рибонуклеотидной последовательности.
В вариантах осуществления первого - четвертого аспектов молекула РНК включает две или более смысловых рибонуклеотидных последовательностей, каждая из которых является идентичной по последовательности области молекулы РНК-мишени, и молекула РНК включает одну или более антисмысловых рибонуклеотидных последовательностей, спаренных со смысловыми рибонуклеотидными последовательностями, где тот или больше антисмысловых последовательностей дополнительны, предпочтительно полностью дополнительны в области целевой молекулы. В варианте осуществления две или более смысловых рибонуклеотидных последовательностей идентичны по последовательности различным областям одной молекулы РНК-мишени, которые могут быть или могут не быть смежными в молекуле РНК-мишени. В варианте осуществления две или более смысловых рибонуклеотидных последовательностей идентичны по последовательности области других молекул РНК-мишеней. В варианте осуществления две или более смысловых рибонуклеотидных последовательностей не содержат никаких промежуточных последовательностей петель, т.е. они являются непрерывными относительно молекулы РНК-мишени.
В предпочтительных вариантах осуществления первого - четвертого аспектов молекула РНК включает две или более антисмысловых рибонуклеотидных последовательностей, при этом смысловые рибонуклеотидные последовательности спарены с ними, и каждая такая антисмысловая последовательность комплементрна области молекулы РНК-мишени. Области молекулы РНК-мишени, к которой они комплементарны, могут быть или могут не быть смежными в молекуле РНК-мишени. В варианте осуществления две или более антисмысловых рибонуклеотидных последовательностей комплементарны разным областям одной молекулы РНК-мишени. В варианте осуществления вторая из двух или более антисмысловых рибонуклеотидных последовательностей комплементарна области другой молекулы РНК-мишени, отличающейся от первой из двух или более антисмысловых рибонуклеотидных последовательностей. В предпочтительном варианте осуществления две или более антисмысловых рибонуклеотидных последовательностей не содержат никаких промежуточных последовательностей петель, т.е. они являются непрерывными относительно комплемента молекулы РНК-мишени. В предпочтительном варианте осуществления одна или обе из двух или более антисмысловых рибонуклеотидных последовательностей и смысловых рибонуклеотидных последовательностей спариваются по всей их полной длине с образованием канонических пар оснований или некоторого числа канонических и некоторого числа неканонических пар оснований, предпочтительно пар оснований G:U.
В предпочтительном варианте осуществления первого - четвертого аспектов молекула РНК является одиночной цепью рибонуклеотидов. Например, молекула РНК может включать одиночную цепь рибонуклеотидов, имеющую 5'-конец, по меньшей мере одну смысловую рибонуклеотидную последовательность, которая имеет длину по меньшей мере 21 нуклеотид, антисмысловую рибонуклеотидную последовательность, которая полностью спарена с каждой смысловой рибонуклеотидной последовательностью на протяжении по меньшей мере 21 последовательного нуклеотида, по меньшей мере две последовательности петель и 3'-конец. В порядке от 5' к 3' может быть расположена смысловая рибонуклеотидная последовательность, а затем антисмысловая рибонуклеотидная последовательность, или наоборот. В варианте осуществления рибонуклеотид на 5'-конце и рибонуклеотид на 3'-конце являются смежными, каждый образует пару оснований и не связан ковалентно напрямую, см., например, Фигуру 1.
В другом варианте осуществления первого - четвертого аспектов молекула РНК включает первую антисмысловую рибонуклеотидную последовательность, которая гибридизуется с первой областью РНК-мишени, вторая антисмысловая рибонуклеотидная последовательность гибридизуется со второй областью РНК-мишени, причем вторая область РНК-мишени отличается от первой области РНК-мишени, и молекула РНК включает только одну смысловую рибонуклеотидную последовательность, которая гибридизуется с РНК-мишенью, где две антисмысловых последовательности не являются смежными в молекуле РНК. В варианте осуществления первая и вторая области РНК-мишени являются смежными в РНК-мишени. В альтернативе они не являются смежными.
В другом варианте осуществления первого - четвертого аспектов молекула РНК включает первую смысловую рибонуклеотидную последовательность, которая по меньшей мере на 60% идентична первой области РНК-мишени, вторую смысловую рибонуклеотидную последовательность, которая по меньшей мере на 60% идентична второй области РНК-мишени, при этом вторая область РНК-мишени отличается от первой области РНК-мишени, и молекула РНК включает только одну антисмысловую рибонуклеотидную последовательность, которая гибридизуется с РНК-мишенью, где две смысловых последовательностей не являются смежными в молекуле РНК. В варианте осуществления первая и вторая области РНК-мишени являются смежными в молекуле РНК-мишени. В альтернативе они не являются смежными. В предпочтительных вариантах осуществления первая и вторая смысловые рибонуклеотидные последовательности независимо являются по меньшей мере на 70%, по меньшей мере на 80%, по меньшей мере на 90%, по меньшей мере на 95% или по меньшей мере на 99% идентичными соответствующей области РНК-мишени, т.е. первая смысловая последовательность может быть по меньшей мере на 70% идентична своей области-мишени, а вторая последовательность - по меньшей мере на 80% идентична своей последовательности-мишени, и т.д.
В предпочтительном варианте осуществления первого - четвертого аспектов молекула РНК является одиночной цепью рибонуклеотидов, имеющей 5'-конец, по меньшей мере одну смысловую рибонуклеотидную последовательность, которая имеет длину по меньшей мере 21 нуклеотид, антисмысловую рибонуклеотидную последовательность, которая полностью спарена с каждой смысловой рибонуклеотидной последовательностью на протяжении по меньшей мере 21 последовательного нуклеотида, по меньшей мере две последовательности петель и 3'-конец. В более предпочтительном варианте осуществления спаривание оснований в молекуле РНК происходит в двухцепочечной области, которая имеет длину по меньшей мере 21 последовательное основание и включает некоторые неканонические пары оснований, наиболее предпочтительно некоторые пары оснований G:U, при этом двухцепочечная область включает по меньшей мере одну смысловую рибонуклеотидную последовательность, которая имеет длину по меньшей мере 21 нуклеотид.
В предпочтительных вариантах осуществления первого и второго аспектов второй РНК-компонент отличается тем, что:
i) вторая смысловая рибонуклеотидная последовательность состоит по меньшей мере из ковалентно связанных 20 последовательных рибонуклеотидов, в порядке от 5' к 3', второго 5'-рибонуклеотида, третьей последовательности РНК и третьего 3'-рибонуклеотида,
ii) вторая антисмысловая рибонуклеотидная последовательность состоит по меньшей мере из ковалентно связанных 20 последовательных рибонуклеотидов, в порядке от 5' к 3', третьего 5'-рибонуклеотида, четвертой последовательности РНК и второго 3'-рибонуклеотида,
iii) второй 5'-рибонуклеотид спаривается со вторым 3'-рибонуклеотидом,
iv) третий 3'-рибонуклеотид спаривается с третьим 5'-рибонуклеотидом,
где химерная молекула РНК способна к процессингу в эукариотической клетке или in vitro, при этом вторая антисмысловая рибонуклеотидная последовательность расщепляется с получением короткой молекулы антисмысловой РНК (асРНК) длиной 20-24 рибонуклеотида. Наиболее предпочтительно молекулы асРНК, полученные из второй антисмысловой последовательности, способны снижать экспрессию РНК-мишени, без или в комбинации с молекулами асРНК, полученными из первой антисмысловой последовательности первого РНК-компонента.
В предпочтительном варианте осуществления четвертого аспекта второй РНК-компонент отличается тем, что:
i) вторая смысловая рибонуклеотидная последовательность состоит по меньшей мере из ковалентно связанных 20 последовательных рибонуклеотидов, в порядке от 5' к 3', третьего 5'-рибонуклеотида, третьей последовательности РНК и третьего 3'-рибонуклеотида,
ii) вторая антисмысловая рибонуклеотидная последовательность состоит по меньшей мере из ковалентно связанных 20 последовательных рибонуклеотидов, в порядке от 5' к 3', четвертого 5'-рибонуклеотида, четвертой последовательности РНК и четвертого 3' рибонуклеотида,
iii) третий 5'-рибонуклеотид спаривается с четвертым 3'-рибонуклеотидом,
iv) третий 3'-рибонуклеотид спаривается с третьим 5'-рибонуклеотидом,
где химерная молекула РНК способна к процессингу в эукариотической клетке или in vitro, при этом вторая антисмысловая рибонуклеотидная последовательность расщепляется с получением короткой молекулы антисмысловой РНК (асРНК) длиной 20-24 рибонуклеотида.
В этих предпочтительных вариантах осуществления более предпочтительно, что от 5% до 40% рибонуклеотидов второй смысловой рибонуклеотидной последовательности и второй антисмысловой рибонуклеотидной последовательности, в общей сложности, спарены с образованием неканонических пар оснований или не спарены, и/или вторая область дцРНК не содержит 20 последовательных канонических пар оснований. Более предпочтительно приблизительно 12%, приблизительно 15%, приблизительно 18%, приблизительно 21%, приблизительно 24% или от 15% до 30%, или еще более предпочтительно от 16% до 25% рибонуклеотидов второй смысловой рибонуклеотидной последовательности и второй антисмысловой рибонуклеотидной последовательности, в общей сложности, спарены с образованием неканонических пар оснований или не спарены. В предпочтительных вариантах осуществления по меньшей мере 50%, по меньшей мере 60%, по меньшей мере 70%, по меньшей мере 80%, по меньшей мере 90%, по меньшей мере 95%, по меньшей мере 97% или 100% неканонических пар оснований во второй области дцРНК являются парами оснований G:U. Наиболее предпочтительно в этих вариантах осуществления:
(a) химерная молекула РНК и/или по меньшей мере некоторые молекулы асРНК способны снижать экспрессию или активность молекулы РНК-мишени в эукариотической клетке, или
(b) вторая антисмысловая рибонуклеотидная последовательность включает последовательность по меньшей мере из 20 последовательных рибонуклеотидов, которая по меньшей мере на 50% идентична по последовательности области комплемента молекулы РНК-мишени, предпочтительно по меньшей мере на 60% идентична, более предпочтительно по меньшей мере на 70% идентична, еще более предпочтительно по меньшей мере на 80% идентична, наиболее предпочтительно по меньшей мере на 90% идентична или на 100% идентична области комплемента молекулы РНК-мишени, или
и (a), и (b).
В варианте осуществления первого - четвертого аспектов молекула РНК включает 5'-лидерную последовательность или 5'-удлиняющую последовательность. В варианте осуществления молекула РНК включает 3'-трейлерную последовательность или 3'-удлиняющую последовательность. В предпочтительном варианте осуществления молекула РНК включает и 5'-лидерную/удлиняющую последовательность, и 3'-трейлерную/удлиняющую последовательность.
В варианте осуществления первого - четвертого аспектов каждый рибонуклеотид молекулы РНК ковалентно связан с двумя другими нуклеотидами, т.е. это - ковалентно замкнутый цикл. Также молекула РНК может быть представлена в форме гантели (Фигура 1), но при этом имеет пропуск или ник в одной части двухцепочечной структуры.
В варианте осуществления первого - четвертого аспектов по меньшей мере одна или все последовательности петель молекулы РНК имеют длину больше 20 нуклеотидов. В предпочтительном варианте осуществления по меньшей мере одна из петель молекулы РНК имеет длину от 4 до 1000 рибонуклеотидов. В более предпочтительном варианте осуществления все петли имеет длину от 4 до 1000 рибонуклеотидов. В более предпочтительном варианте осуществления по меньшей мере одна из петель молекулы РНК имеет длину от 4 до 200 рибонуклеотидов. В еще более предпочтительном варианте осуществления все петли имеет длину от 4 до 200 рибонуклеотидов. В еще более предпочтительном варианте осуществления по меньшей мере одна из петель молекулы РНК имеет длину от 4 до 50 рибонуклеотидов. В самом предпочтительном варианте осуществления все петли имеет длину от 4 до 50 рибонуклеотидов. В варианте осуществления эукариотическая клетка является клеткой позвоночного, и каждая петля молекулы РНК имеет длину от 20 до 50 или от 20 до 30 рибонуклеотидов.
В вариантах осуществления первого - четвертого аспектов молекула РНК не имеет ни одного или имеет одно или два, или больше выпетливаний в двухцепочечной области. В этом контексте выпетливание является нуклеотидом или двумя или более смежными нуклеотидами в смысловой или антисмысловой рибонуклеотидной последовательности, которые не спарены в области дцРНК и которые не имеют несовпадающего нуклеотида в соответствующем положении в комплементарной последовательности в области дцРНК. Область дцРНК молекулы РНК может включать последовательность больше 2 или 3 нуклеотидов в смысловой или антисмысловой последовательности или в обеих, которая образует выпетливание из области дцРНК при формировании структуры дцРНК. Последовательность, которая образует выпетливание, может сама формировать некоторое внутреннее спаривание оснований, например, она сама может образовывать структуру стебель-петли.
В вариантах осуществления первого - четвертого аспектов молекула РНК не имеет ни одного или имеет одно или два, или больше выпетливаний в двухцепочечной области. В этом контексте выпетливание является нуклеотидом или двумя или более смежными нуклеотидами в смысловой или антисмысловой рибонуклеотидной последовательности, которые не спарены в области дцРНК и не имеют несовпадающего нуклеотида в соответствующем положении в комплементарной последовательности в области дцРНК. Область дцРНК молекулы РНК может включать последовательность больше чем из 2 или 3 нуклеотидов в смысловой или антисмысловой последовательности, или в обеих, которая образует выпетливание из области дцРНК при формировании структуры дцРНК. Последовательность, образующая выпетливание, может сама формировать некоторое внутреннее спаривание оснований, например, она сама может образовывать структуру стебель-петли.
В варианте осуществления молекула РНК имеет три, четыре или больше петель. В предпочтительном варианте осуществления молекула РНК имеет только две петли. В варианте осуществления первая двухцепочечная область или первая и вторая области дцРНК молекулы РНК включают один или два, или больше нуклеотидов, которые не спарены в двухцепочечной области, или до 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9% или 10% нуклеотидов в двухцепочечной области, которые не спарены.
В предпочтительных вариантах осуществления первого - четвертого аспектов молекула РНК-мишени или молекула РНК согласно изобретению, или обе, находятся в эукариотической клетке. Например, эукариотическая клетка может быть клеткой растения, клеткой животного или клеткой гриба. В варианте осуществления эукариотическая клетка является клеткой гриба. В варианте осуществления молекула РНК согласно изобретению продуцируется в клетке, такой как, например, бактериальная клетка или другая микробная клетка, которая отличается от клетки, включающей РНК-мишень. Аналогичным образом, в варианте осуществления молекула РНК согласно изобретению продуцируется в эукариотической клетке, которая не включает РНК-мишень, когда продуцируется молекула РНК согласно изобретению, но эукариотическая клетка, включающая молекулу РНК согласно изобретению и/или ее процессированные продукты РНК, может стать хозяином РНК-мишени, например, если РНК-мишень является вирусной РНК или другой введенной РНК. Такие клетки могут быть профилактически защищены против вирусной или другой введенной РНК.
В предпочтительных вариантах осуществления первого - четвертого аспектов молекула РНК может быть получена ферментативно при транскрипции in vitro и/или в клетке. В варианте осуществления молекула РНК настоящего изобретения экспрессируется в клетке, т.е. продуцируется в клетке при транскрипции с одной или более нуклеиновых кислот, кодирующих молекулу РНК. Одна или более нуклеиновых кислот, кодирующих молекулу РНК, предпочтительно являются молекулой ДНК, которая может присутствовать в векторе в клетке или может быть интегрирована в геном клетки или ядерный геном клетки, или в пластидную ДНК клетки. Одна или более нуклеиновых кислот, кодирующих молекулу РНК, также могут быть молекулой РНК, такой как вирусный вектор.
Таким образом, в варианте осуществления настоящего изобретения предложена клетка, включающая молекулу РНК, описанную в настоящем документе. В предпочтительном варианте осуществления настоящего изобретения предложена молекула РНК, описанная в настоящем документе, которая экспресировалась в клетке и которая была выделена и/или очищена из клетки. В настоящем изобретении, таким образом, предложен препарат выделенных молекул РНК согласно одному или более из первого - четвертого аспектов, подходящий для введения в клетку, включающую РНК-мишень или потенциально включающую РНК-мишень.
В варианте осуществления одна или более РНК-мишеней кодируют белок. В альтернативе одна или более РНК-мишеней не кодируют белок, такой как рРНК, тРНК, мякРНК или миРНК.
В вариантах осуществления первого - четвертого аспектов приблизительно 12%, приблизительно 15%, приблизительно 18%, приблизительно 21%, приблизительно 24% или от приблизительно 15% до приблизительно 30%, или предпочтительно от приблизительно 16% до приблизительно 25% рибонуклеотидов смысловой рибонуклеотидной последовательности и антисмысловой рибонуклеотидной последовательности, в общей сложности, которые образуют область дцРНК, спарены с образованием неканонической пары оснований или не спарены. В предпочтительном варианте осуществления по меньшей мере 50%, по меньшей мере 60%, по меньшей мере 70%, по меньшей мере 80%, по меньшей мере 90%, по меньшей мере 95%, по меньшей мере 97% или 100% неканонических пар оснований в области дцРНК или во всех областях дцРНК в молекуле РНК являются парами оснований G:U. Нуклеотид G в каждой паре оснований G:U может быть независимо в смысловой рибонуклеотидной последовательности или предпочтительно в антисмысловой рибонуклеотидной последовательности. В отношении нуклеотидов G в парах оснований G:U области дцРНК, предпочтительно по меньшей мере 50% находятся в антисмысловой рибонуклеотидной последовательности, более предпочтительно по меньшей мере 60% или 70%, еще более предпочтительно по меньшей мере 80% или 90% и наиболее предпочтительно по меньшей мере 95% из них находятся в антисмысловой рибонуклеотидной последовательности в области дцРНК. Эта особенность может относиться ко всем областям дцРНК в молекуле РНК. В варианте осуществления меньше 25%, меньше 20%, меньше 15%, меньше 10%, предпочтительно меньше 5%, более предпочтительно меньше 1% или наиболее предпочтительно ни один из рибонуклеотидов в области дцРНК или во всех областях дцРНК в молекуле РНК, в общей сложности, не спарены. В предпочтительном варианте осуществления все из четырех - все из шести рибонуклеотидов в области дцРНК или во всех областях дцРНК образуют неканоническую пару оснований или не спарены в молекуле РНК. В предпочтительном варианте осуществления область дцРНК или все области дцРНК не включают 8 последовательных канонических пар оснований. В альтернативном варианте осуществления область дцРНК включает по меньшей мере 8 последовательных канонических пар оснований, например 8-12 или 8-14 последовательных канонических пар оснований. В предпочтительном варианте осуществления все рибонуклеотиды в области дцРНК спарены с канонической парой оснований или неканонической парой оснований. В варианте осуществления один или более рибонуклеотидов смысловой рибонуклеотидной последовательности и/или один или более рибонуклеотидов антисмысловой рибонуклеотидной последовательности не спарены. В варианте осуществления один или более рибонуклеотидов каждой смысловой рибонуклеотидной последовательности и один или более рибонуклеотидов каждой антисмысловой рибонуклеотидной последовательности не спарены в молекуле РНК согласно изобретению.
В варианте осуществления антисмысловая последовательность РНК первого РНК-компонента или второго РНК-компонента, или обоих, меньше чем на 100% идентична или на приблизительно от 80% до 99,9% идентична, или на приблизительно от 90% до 98% идентична, или на приблизительно от 95% до 98% идентична, предпочтительно на 98-99,9% идентична по последовательности комплементу области молекулы РНК-мишени или двум таким областям, которые могут быть или могут не быть смежными в молекуле РНК-мишени. В предпочтительном варианте осуществления антисмысловая последовательность РНК на 100% идентична по последовательности области комплемента молекулы РНК-мишени, например, области, включающей 21, 23, 25, 27, 30 или 32 последовательных нуклеотида. В варианте осуществления смысловая или антисмысловая рибонуклеотидная последовательность, или обе, имеют длину по меньшей мере 40, по меньшей мере 50, по меньшей мере приблизительно 100, по меньшей мере приблизительно 200, по меньшей мере приблизительно 300, по меньшей мере приблизительно 400, по меньшей мере приблизительно 500, по меньшей мере приблизительно 600, по меньшей мере приблизительно 700, по меньшей мере приблизительно 800, по меньшей мере приблизительно 900, по меньшей мере приблизительно 1000 или от приблизительно 100 до приблизительно 1000 последовательных нуклеотидов. Длина по меньшей мере 100 нуклеотидов предпочтительна при использовании молекулы РНК в клетках растений или клетках грибов или в случае клеток беспозвоночных животных. Длина смысловой и антисмысловой рибонуклеотидной последовательности в дцРНК 50 нуклеотидов или меньше, например 31-50 нуклеотидов, предпочтительна при использовании молекулы РНК в клетках позвоночных животных. В варианте осуществления количество рибонуклеотидов в смысловой рибонуклеотидной последовательности составляет от приблизительно 90% до приблизительно 110%, предпочтительно от 95% до 105%, более предпочтительно от 98% до 102%, еще более предпочтительно от 99% до 101% от количества рибонуклеотидов в антисмысловой рибонуклеотидной последовательности. В наиболее предпочтительном варианте осуществления количество рибонуклеотидов в смысловой рибонуклеотидной последовательности совпадает с количеством рибонуклеотидов в антисмысловой рибонуклеотидной последовательности. Эти признаки могут быть применены к каждой области дцРНК в молекуле РНК.
В вариантах осуществления первого - четвертого аспектов первый 3'-рибонуклеотид и второй 5'-рибонуклеотид в молекуле РНК ковалентно соединены последовательностью петли, состоящей по меньшей мере из 4 рибонуклеотидов или от 4 до 1000 рибонуклеотидов или предпочтительно от 4 до 200 рибонуклеотидов, более предпочтительно от 4 до 50 рибонуклеотидов. В варианте осуществления молекула РНК дополнительно включает 5'-удлиняющую последовательность, ковалентно связанную с первым 5'-рибонуклеотидом, или 3'-удлиняющую последовательность, ковалентно связанную со вторым 3'-рибонуклеотидом, или обе. В варианте осуществления химерная молекула РНК дополнительно включает 5'-удлиняющую последовательность, ковалентно связанную со вторым 5'-рибонуклеотидом, или 3'-удлиняющую последовательность, ковалентно связанную с первым 3'-рибонуклеотидом, или обе. В этом варианте осуществления молекула РНК включает две отдельных цепи РНК, которые гибридизуются с образованием молекулы РНК, хотя она может быть синтезирована при транскрипции с молекулы нуклеиновой кислоты в виде одного транскрипта РНК и затем процессирована с включением двух цепей РНК.
Полная длина молекулы РНК согласно изобретению, продуцируемая в виде одной цепи РНК, после сплайсинга каких-либо интронов, но перед каким-либо процессингом молекулы РНК ферментами Dicer или другими РНКазами, как правило, имеют длину от 50 до 2000 рибонуклеотидов, предпочтительно от 60 или 70 до 2000 рибонуклеотидов, более предпочтительно от 80 или 90 до 2000 рибонуклеотидов, еще более предпочтительно от 100 или 110 до 2000 рибонуклеотидов. В предпочтительных вариантах осуществления минимальная длина молекулы РНК составляет 120, 130, 140, 150, 160, 180 или 200 нуклеотидов, а максимальная длина составляет 400, 500, 600, 700, 800, 900, 1000, 1200, 1400, 1500 или 2000 рибонуклеотидов. Рассматривается каждая комбинация указанных минимальных и максимальных длин. Продукция молекул РНК такой длины с готовностью достигается при транскрипции in vitro или в клетках, таких как бактериальные или другие микробные клетки, или в эукариотической клетке, в которой ген-мишень необходимо даунрегулировать.
В варианте осуществления первого - четвертого аспектов химерная молекула РНК включает две или более областей дцРНК, которые являются одинаковыми или, предпочтительно, разными.
В предпочтительных вариантах осуществления первого - четвертого аспектов молекула РНК экспрессируется в эукариотической клетке, т.е. продуцируется в клетке в результате транскрипции. В этих вариантах осуществления большая часть молекул дцРНК образуется при процессинге молекулы РНК, которая имеет длину 22 и/или 20 рибонуклеотидов, по сравнению с процессингом аналогичной молекулы РНК, имеющей соответствующую область дцРНК, которая полностью спарена с каноническими парами оснований. Таким образом, молекулы РНК таких вариантов осуществления с большей легкостью процессируются с получением коротких антисмысловых РНК длиной 22 и/или 20 рибонуклеотидов, чем аналогичная молекула РНК, область дцРНК которой полностью спарена с каноническими парами оснований, в качестве части от общего количества асРНК длиной 20-24 нуклеотидов, полученных из молекулы РНК.
В варианте осуществления молекула РНК согласно изобретению включает комбинацию двух или более отличительных признаков молекулы РНК, описанной в настоящем документе.
В другом аспекте настоящего изобретения предложен полинуклеотид, кодирующий молекулу РНК, описанную в настоящем документе, предпочтительно химерную молекулу РНК, описанную в настоящем документе. В варианте осуществления полинуклеотид является конструкцией ДНК, которая может быть интегрирована в более крупную молекулу ДНК, такую как хромосома. В варианте осуществления полинуклеотид функционально связан с промотором, способным направлять экспрессию молекулы РНК в клетке-хозяине. Клетка-хозяин может быть бактериальной клеткой, такой как E. coli, клетка гриба, такая как дрожжевая клетка, или эукариотическая клетка, такая как клетка растения или клетка животного. В варианте осуществления промотор является гетерологичным по отношению к полинуклеотиду. Полинуклеотид, кодирующий молекулу РНК, может быть химерным или рекомбинантным полинуклеотидом, или выделенным и/или экзогенным полинуклеотидом. В варианте осуществления промотор может функционировать in vitro, например, промотор бактериофага, такой как промотор РНК-полимеразы T7 или промотор РНК-полимеразы SP6. В варианте осуществления промотор является промотором РНК-полимеразы III, таким как промотор U6 или промотор H1. В варианте осуществления промотор является промотором РНК-полимеразы II, который может быть конститутивным промотором, тканеспецифическим промотором, регулируемым развитием промотором или индуцируемым промотором. В варианте осуществления полинуклеотид кодирует молекулу РНК-предшественника, включающую интрон по меньшей мере в одной последовательности петли, который может быть вырезан во время или после транскрипции полинуклеотида в клетке-хозяине. В варианте осуществления настоящего изобретения предложен вектор, включающий полинуклеотид, описанный в настоящем документе. В варианте осуществления вектор является вирусным вектором. В варианте осуществления вектор является плазмидным вектором, таким как бинарный вектор, подходящий для применения с Agrobacterium tumefaciens.
В варианте осуществления, где полинуклеотид или вектор согласно изобретению находятся в эукариотической клетке-хозяине, предпочтительно в растении, промоторная область полинуклеотида или вектора, функционально связанного с областью, кодирующей молекулу РНК согласно изобретению, имеет более низкий уровень метилирования по сравнению с промотором соответствующего полинуклеотида или вектора, кодирующего молекулу РНК, имеющую соответствующую область дцРНК, которая полностью спарена с каноническими парами оснований. В варианте осуществления более низкий уровень метилирования составляет меньше 50%, меньше 40%, меньше 30% или меньше 20% по сравнению с промотором соответствующего полинуклеотида или вектора. В варианте осуществления клетка-хозяин включает по меньшей мере две копии полинуклеотида или вектора, кодирующего молекулу РНК согласно изобретению. В этом варианте осуществления:
i) уровень снижения экспрессии и/или активности молекулы РНК-мишени в эукариотической клетке является по меньшей мере таким же по сравнению с соответствующей эукариотической клеткой, содержащей одну копию полинуклеотида или вектора, и/или
ii) уровень снижения экспрессии и/или активности молекулы РНК-мишени в эукариотической клетке является более низким когда по сравнению с соответствующей клеткой, включающей молекулу РНК, имеющую соответствующую область дцРНК, которая полностью спарена с каноническими парами оснований.
В другом аспекте настоящего изобретения предложена клетка-хозяин, включающая молекулу РНК, описанную в настоящем документе, полинуклеотид, описанный в настоящем документе, или вектор, включающий их. В варианте осуществления клетка-хозяин является нечеловеческой клеткой, такой как клетка бактерии, клетка гриба, клетка растения или клетка не относящегося к человеку животного. В варианте осуществления клетка является нечеловеческой клеткой или клеткой человека в клеточной культуре. В варианте осуществления клетка является эукариотической клеткой, такой как клетка, отличающаяся от клетки животного. В варианте осуществления клетка является микробной клеткой, такой как прокариотическая клетка. В варианте осуществления клетка-хозяин является живой. В альтернативном варианте осуществления клетка-хозяин является мертвой.
В другом аспекте настоящего изобретения предложен не относящийся к человеку организм, включающий молекулу РНК согласно изобретению, предпочтительно химерную молекулу РНК, описанную в настоящем документе, полинуклеотид или вектор согласно изобретению, включающий их, или клетка-хозяин, включающая их. В варианте осуществления не относящийся к человеку организм является трансгенным, поскольку он включает полинуклеотид согласно изобретению. В варианте осуществления полинуклеотид стабильно интегрирован в геном не относящегося к человеку организма.
В другом аспекте настоящего изобретения предложен способ получения молекулы РНК согласно изобретению, включающий экспрессию полинуклеотида согласно изобретению в клетке-хозяине или бесклеточной системе экспрессии. В этом варианте осуществления способ может дополнительно включать, по меньшей мере, частичную очистку молекулы РНК, или нет.
В другом аспекте настоящего изобретения предложен способ получения клетки или не относящегося к человеку организма, предпочтительно растения или гриба, включающий введение полинуклеотида или вектора согласно изобретению в клетку, предпочтительно клетку растения или гриба, предпочтительно так, чтобы полинуклеотид или вектор или его часть, кодирующая молекулу РНК, были стабильно интегрированы в геном клетки. В варианте осуществления не относящийся к человеку организм создан из клетки или дочерней клетки, например, путем регенерации растения. В варианте осуществления не относящийся к человеку организм создан путем введения клетки или одной или более дочерних клеток в не относящийся к человеку организм. Альтернативно стабильной интеграции полинуклеотида или вектора в геном клетки, полинуклеотид или вектор могут быть введены в клетку без интеграции полинуклеотида или вектора в геном, например, для транзиентной экспрессии молекулы РНК в клетке или организме.
В другом аспекте настоящего изобретения предложен экстракт клетки-хозяина или организма или их части согласно изобретения, где экстракт включает молекулу РНК согласно изобретению, малые молекулы РНК (длиной 20-24 нуклеотида), полученные в результате процессинга молекулы РНК, или и то, и другое, и/или полинуклеотид или вектор согласно изобретению. В варианте осуществления настоящего изобретения предложена композиция, включающая одну или более молекул РНК согласно изобретению, малые молекулы РНК (длиной 20-24 нуклеотида), полученные в результате процессинга молекулы РНК, или и то, и другое, полинуклеотид согласно изобретению, вектор согласно изобретению, клетка-хозяин согласно изобретению или экстракт, полученный способом согласно изобретению, и один или более подходящих носителей. В варианте осуществления композиция является фармацевтической композицией, такой как композиция, подходящая для введения человеку или другому животному. Фармацевтическая композиция может подходить для профилактики или лечения заболевания или для наружного применения, такого как косметическое применение. В варианте осуществления композиция подходит для нанесения на растение, предпочтительно растение или популяцию растений в полевых условиях, или на насекомое или популяцию насекомых. В варианте осуществления композиция подходит для нанесения на сельскохозяйственную культуру, например, путем распыления на культурные растения в полевых условиях.
В варианте осуществления экстракт или композиция, включающие молекулу РНК согласно изобретению или малые молекулы РНК (длиной 20-24 нуклеотида), полученные в результате процессинга молекулы РНК, или и то, и другое, дополнительно включают по меньшей мере одно соединение, которое повышает стабильность молекулы РНК или полинуклеотида и/или вектора, при этом по меньшей мере одно соединение облегчает захват молекулы РНК, полинуклеотида или вектора клеткой, такой как, например, клетка организма. В варианте осуществления соединение является агентом, способствующим трансфекции, например, липидсодержащим соединением.
В другом аспекте настоящего изобретения предложен способ снижения или даунрегуляции уровня и/или активности молекулы РНК-мишени в клетке или организме, включающий введение в клетку или организм одной или более молекул РНК согласно изобретению или малых молекул РНК (длиной 20-24 нуклеотида), полученных в результате процессинга молекулы РНК, или и то, и другое, полинуклеотида согласно изобретению, вектора согласно изобретению или композиции согласно изобретению. В варианте осуществления молекула РНК-мишени кодирует белок. В варианте осуществления способ снижает уровень и/или активность больше чем одной молекулы РНК-мишени, причем молекулы РНК-мишени являются различными, например, снижается уровень и/или активность двух или более РНК-мишеней, родственных по последовательности, например, из семейства генов.
В другом аспекте настоящего изобретения предложен способ контроля не относящегося к человеку организма, включающий доставку в не относящийся к человеку организм одной или более молекул РНК согласно изобретению или малых молекул РНК (длиной 20-24 нуклеотида), полученных путем процессинга молекулы РНК, или и то, и другое, или полинуклеотида или вектора согласно изобретению, клетки-хозяина согласно изобретению, экстракта, полученного способом согласно изобретению, или композиции согласно изобретению, где молекула РНК или малые молекулы РНК оказывают токсическое воздействие на не относящийся к человеку организм. В варианте осуществления не относящийся к человеку организм является членистоногим, таким как, например, насекомое, или растением таким как, например, сорняк. В варианте осуществления не относящийся к человеку организм является растением, при этом членистоногое поедает такое растение или его часть, посредством чего осуществляется контроль членистоногого.
В варианте осуществления настоящего изобретения предложен способ предупреждения или лечения заболевания у субъекта, включающий введение субъекту одной или более молекул РНК согласно изобретению или малых молекул РНК (длиной 20-24 нуклеотида), полученных путем процессинга молекулы РНК, или и того, и другого, полинуклеотида или вектора согласно изобретению, клетки-хозяина согласно изобретению, экстракта, полученного способом согласно изобретению, или композиции согласно изобретению, где молекула РНК или малые молекулы РНК оказывают благоприятный эффект по меньшей мере в отношении одного симптома заболевания. В варианте осуществления молекула РНК или малые молекулы РНК, полинуклеотид, вектор или композицию применяют местно, перорально или путем инъекции. В варианте осуществления субъект является позвоночным животным. В варианте осуществления позвоночное животное является млекопитающим, таким как человек, сельскохозяйственное животное, такое как рогатый скот или овцы, или птицы, такие как куры и другая домашняя птица.
В другом аспекте настоящего изобретения предложена молекула РНК согласно изобретению, полинуклеотид или вектор согласно изобретению, клетка-хозяин согласно изобретению, экстракт, полученный способом согласно изобретению, или композиция согласно изобретению для применения при лечении заболевания у субъекта, где молекула РНК или малые молекулы РНК оказывают благоприятный эффект в отношении по меньшей мере одного симптома заболевания. В варианте осуществления настоящего изобретения предложено применение молекулы РНК согласно изобретению или малых молекул РНК, полученных из нее, полинуклеотида или вектора согласно изобретению, клетки-хозяина согласно изобретению, экстракта, полученного способом согласно изобретению, или композиции согласно изобретению для производства лекарственного средства для предупреждения или лечения заболевания у субъекта, где молекула РНК или малые молекулы РНК, полученные из нее, оказывают благоприятный эффект в отношении по меньшей мере одного симптома заболевания.
В другом аспекте настоящего изобретения предложен набор, включающий одну или более молекул РНК согласно изобретению или малых молекул РНК, полученных из нее, полинуклеотид или вектор согласно изобретению, клетку-хозяина согласно изобретению, экстракт, полученный способом согласно изобретению, или композицию согласно изобретению.
Любой вариант осуществления в настоящем документе должен применяться с соответствующими изменениями к любому другому варианту осуществления, если специально не указано иное.
Настоящее изобретение не должно быть ограничено в объеме определенными вариантами осуществления, описанными в настоящем документе, которые предназначены лишь в целях иллюстрации. Функционально эквивалентные продукты, композиции и способы в явной форме включены в рамки изобретения, как описано в настоящем документе.
По всему тексту настоящего описания, если прямо не указано иное или иное не следует из контекста, ссылка на один этап, композицию, группу этапов или группу композиций должна охватывать один и множество (т.е. один или больше) таких этапов, композиций, групп этапов или групп композиций.
Далее изобретение описано посредством следующих неограничивающих Примеров и со ссылкой на сопровождающие фигуры.
КРАТКОЕ ОПИСАНИЕ СОПРОВОЖДАЮЩИХ ЧЕРТЕЖЕЙ
Фигура 1. Схематические конструкции двух молекул кпРНК. (A) Эта молекула кпРНК включает смысловую последовательность, которую можно рассматривать как две смежные смысловые последовательности, ковалентно связанные без промежуточной спейсерной последовательности и обладающие идентичностью с РНК-мишенью, антисмысловую последовательность, которая комплементарна смысловой последовательности и которая разделена на две области - 5'-область и 3'-область, и две петли, которые отделяют смысловую от антисмысловой последовательности. (B) Эта молекула кпРНК содержит антисмысловую последовательность, которую можно рассматривать как две смежные антисмысловые последовательности, ковалентно связанные без промежуточной спейсерной последовательности и обладающие идентичностью с комплементом РНК-мишени, смысловую последовательность, которая комплементарна антисмысловой последовательности и которая разделена на две области, и две петли, которые отделяют смысловую от антисмысловой последовательности. Молекула РНК, полученная путем транскрипции, например, путем транскрипции in vitro с промотора, такого как промотор T7 или Sp6, отжигается на себя при спаривании оснований между комплементарной смысловой и антисмысловой последовательностями с образованием двухцепочечной области с петлей на каждом конце и имеет "ник" в антисмысловой или смысловой последовательности. Дополнительные последовательности могут быть связаны с 5'- и/или 3'-концами как 5'- или 3'-удлинители.
Фигура 2. кпРНК более эффективна при формировании дцРНК, чем отжиг смысловой/антисмысловой шпилечной РНК. Показаны схематические представления трех форм двухцепочечных молекул РНК: A, обычная дцРНК, образованная при отжиге двух отдельных цепей; B, шпилечная РНК, имеющая 5'- и 3'-удлинитель; и C, молекула кпРНК. На нижней панели показан фотоснимок после гель-электрофореза РНК транскриптов трех типов молекул РНК, направленно воздействующих на ген GUS или на ген GFP.
Фигура 3. Нозерн-блот гибридизация обработанных (A и B) и необработанных дистальных (C и D) тканей показывает, что кпРНК более стабильна, чем дцРНК, и распространяется в ткани табачного листа. В дистальных тканях (C и D, верхняя панель) сигнал дцРНК не был обнаружен, в отличие от мощных сигналов кпРНК.
Фигура 4. Обработка кпРНК вызывала даунрегуляцию GUS как в обработанной области (1), так и в необработанной области выше (3).
Фигура 5. кпРНК вызывает сайленсинг гена FAD2.1 в листьях N. benthamiana.
Фигура 6. Нозерн-блот гибридизация подтверждает сильную даунрегуляцию мРНК FAD2.1 при обработке ledFAD2.1 через 6 и 24 часа.
Фигура 7. Выравнивание нуклеотидных последовательностей области гена-мишени GUS (SEQ ID NO:14) и смысловой последовательности конструкции hpGUS[G:U] (нуклеотиды 9-208 из SEQ ID NO:11). 52 цитидин-нуклеотида (C) заменены тимидин-нуклеотидами (T). Консервативные нуклеотиды отмечены звездочкой, замененные C не отмечены звездочкой.
Фигура 8. Выравнивание нуклеотидных последовательностей области гена-мишени GUS (SEQ ID NO:14) и смысловой последовательности конструкции hpGUS[1:4] (нуклеотиды 9-208 из SEQ ID NO:12). Каждый 4-й нуклеотид в hpGUS[1:4] был заменен в сравнении с соответствующей смысловой последовательностью дикого типа, в результате для каждого 4-го нуклеотида, C был изменен на G, G был изменен на C, A был изменен на T, и T был изменен на A. Консервативные нуклеотиды отмечены звездочкой, замененные G и C не отмечены звездочкой, замененные A и Т отмечены двоеточием.
Фигура 9. Выравнивание нуклеотидных последовательностей области гена-мишени GUS (SEQ ID NO:14) и смысловой последовательности конструкции hpGUS[2:10] (нуклеотиды 9-208 из SEQ ID NO:13). Каждый 9-й и 10-й нуклеотиды в каждом блоке из 10 нуклеотидов в hpGUS[2:10] заменены в сравнении с соответствующей смысловой последовательностью дикого типа, в результате для каждого 9-го и 10-го нуклеотида, C был изменен на G, G был изменен на C, A был изменен на T, и T был изменен на A. Консервативные нуклеотиды отмечены звездочкой, замененные G и C не отмечены звездочкой, замененные A и Т отмечены двоеточием.
Фигура 10. Схематическая диаграмма, на которой показаны структуры генетических конструкций, кодирующих модифицированные шпилечные РНК, направленно воздействующие на мРНК GUS.
Фигура 11. Схематическая диаграмма вектора pWBPPGH, используемого для трансформации растений табака, обеспечивающая ген-мишень GUS. T-ДНК идет от правого бордера (RB) до левого бордера (LB) вектора. Ген селективного маркера на T-ДНК является геном 35S-HPT-tm1', кодирующим устойчивость к гигромицину.
Фигура 12. Активность GUS в растениях, трансформированных конструкциями, кодирующими модифицированные шпилечные РНК для снижения экспрессии ген-мишени GUS. Без hp: контрольные растения PPGH11 и PPGH24 без конструкций hpGUS. Количество растений, демонстрирующих меньше чем 10% активность GUS по сравнению с соответствующими контрольными растениями PPGH11 или PPGH24, и процент таких растений относительно числа протестированных растений приведены в скобках.
Фигура 13. (A) Средняя активность GUS всех трансгенных растений: 59 растений hpGUS[wt], 74 - hpGUS[G:U], 33 - hpGUS[1:4] и 41 - hpGUS[2:10]. (B) Средняя активность GUS всех растений после сайленсинга (32 - hpGUS[wt], 71 - hpGUS[G:U], 33 - hpGUS[1:4] и 28 - hpGUS[2:10].
Фигура 14. Активность GUS потомства трансгенных растений, содержащих hpGUS[wt], hpGUS[G:U] или hpGUS[1:4].
Фигура 15. Авторадиограмма Саузерн-блоттинга ДНК из 16 растений, трансформированных конструкцией hpGUS[G:U]. ДНК расщепляли HindIII перед гель-электрофорезом и метили зондом OCS-T. Дорожка 1: маркеры мол.веса (HindIII-расщепленная ДНК лямбда); Дорожки 2 и 3 - ДНК родительских растений PPGH11 и PPGH24; Дорожки 4-19: ДНК из 16 различных трансгенных растений.
Фигура 16. Ауторадиограмма эксперимента Нозерн-блот гибридизации с целью обнаружения смысловых (верхняя панель) и антисмысловых (более низкая группа) кРНК, полученных из шпилечных РНК, экспрессируемых в трансгенном табаке. Дорожки 1 и 2 содержали РНК, полученную из родительских растений PPGH11 и PPGH24, не содержащих конструкции hpGUS. Дорожки 3-11 содержали РНК из растений hpGUS[wt], и дорожки 12-20 содержали РНК из растений hpGUS[G:U].
Фигура 17. Авторадиограмма Нозерн-блот гибридизации с целью обнаружения антисмысловых кРНК из трансгенных растений. Дорожки 1-10 из растений hpGUS[wt], дорожки 11-19 из растений hpGUS[G:U]. Антисмысловые кРНК имеют подвижность, соответствующую длине 20-24 нуклеотида. Блот повторно метили антисмысловыми к РНК U6 в качестве контроля нанесения на дорожку.
Фигура 18. Авторадиограмма повторной Нозерн-блот гибридизации с целью обнаружения антисмысловых кРНК из трансгенных растений
Фигура 19. Анализ метилирования ДНК области соединения 35S промотора и смысловой области GUS в конструкциях hpGUS в трансгенных растениях. Фрагменты соединения амплифицировали с помощью ПЦР с (+) или без (-) предварительной обработки растительной ДНК ферментом McrBC.
Фигура 20. Анализ метилирования ДНК области 35S промотора в конструкциях hpGUS в трансгенных растениях. Фрагменты 35S амплифицировали с помощью ПЦР с (+) или без (-)предварительной обработки растительной ДНК ферментом McrBC.
Фигура 21. Распределение по размеру и представленность процессированной РНК. (A) конструкции EIN2. (B) конструкции GUS.
Фигура 22. Выравнивание смысловой последовательности (верхняя последовательность, нуклеотиды 17-216 из SEQ ID NO:22) конструкции hpEIN2[G:U] и нуклеотидной последовательности (нижняя последовательность, SEQ ID NO:27) области кДНК, соответствующей гену-мишени EIN2 A. thaliana. Смысловая последовательность была получена путем замены 43 цитидин-нуклеотидов (C) в последовательности дикого типа тимидин-нуклеотидами (T). Консервативные нуклеотиды отмечены звездочкой, замененные C не отмечены звездочкой.
Фигура 23. Выравнивание смысловой последовательности (верхняя последовательность, нуклеотиды 13-212 из SEQ ID NO:24) конструкции hpCHS[G:U] с нуклеотидной последовательностью области кДНК, соответствующей гену-мишени CHS A. thaliana (SEQ ID NO:28, нижняя последовательность). Смысловая последовательность была получена путем замены 65 цитидин-нуклеотидов (C) в последовательности дикого типа тимидин-нуклеотидами (T). Консервативные нуклеотиды отмечены звездочкой, замененные C не отмечены звездочкой.
Фигура 24. Выравнивание антисмысловой последовательности (верхняя последовательность, нуклеотиды 8-207 из SEQ ID NO:25) конструкции hpEIN2[G:U/U:G] и нуклеотидной последовательности (нижняя последовательность, SEQ ID NO:29) области комплемента гена-мишени EIN2 A. thaliana. Антисмысловая последовательность была получена путем замены 49 цитидин-нуклеотидов (C) в последовательности дикого типа тимидин-нуклеотидами (T). Консервативные нуклеотиды отмечены звездочкой, замененные C не отмечены звездочкой.
Фигура 25. Выравнивание антисмысловой последовательности (верхняя последовательность, нуклеотиды 13-212 из SEQ ID NO:26) конструкции hpCHS[G:U/U:G] и нуклеотидной последовательности (нижняя последовательность, SEQ ID NO:30) области комплемента гена-мишени CHS A. thaliana. Антисмысловая последовательность была получена путем замены 49 цитидин-нуклеотидов (C) в последовательности дикого типа тимидин-нуклеотидами (T). Консервативные нуклеотиды отмечены звездочкой, замененные C не отмечены звездочкой.
Фигура 26. Схематические диаграммы конструкций шРНК с этилен-нечувствительным 2 (EIN2) и халконсинтазой (CHS). 35S: 35S промотор CaMV; области EIN2 и CHS показаны в виде последовательности дикого типа (wt) или G:U-модифицированной последовательности (G:U). Стрелки указывают ориентацию фрагментов ДНК - стрелки, направленные справа налево, указывают антисмысловые последовательности. Также показаны сайты рестрикции.
Фигура 27. Длина гипокотиля рассады трансгенных растений A. thaliana в анализе EIN2, содержащих hpEIN2[wt] или hpEIN2[G:U]
Фигура 28. кОТ-ПЦР для мРНК CHS в трансгенных A. thaliana, трансгенных по конструкции hpCHS[wt] или hpCHS[G:U], нормализованные по уровням РНК актина-2. Col-0 - A. thaliana дикого типа (нетрансгенный).
Фигура 29. Авторадиограмма Нозерн-блот гибридизации РНК из растений, трансформированных hpEIN2[wt] или hpEIN2[G:U]. На верхней панели показана длина гипокотиля для линий. Авторадиограмма показывает Нозерн-блот, меченный смысловым зондом EIN2 для обнаружения антисмысловых кРНК. Тот же блот повторно метили РНК зондом U6 в качестве контроля нанесения (РНК U6).
Фигура 30. Анализ метилирования ДНК 35S промотора и 35S-смысловых EIN2 последовательностей в геномной ДНК трансгенных растений A. thaliana.
Фигура 31. Уровни метилирования ДНК в промоторе и 5'-области конструкций шпилечной РНК.
Фигура 32. 35S промотор в последних метилированных линиях популяции hpEIN2[wt] все еще показывает значительное метилирование.
Фигура 33. 35S промотор в линиях G:U hpEIN2 показывает лишь слабое метилирование (<10%).
Фигура 34. Сайленсинг гена под действием кпРНК и шРНК с G:U в клетках CHO и Vero через 72 часа.
Фигура 35. Гантельные плазмиды, протестированные в клетках Hela через 48 часов.
Фигура 36. Примеры возможных модификаций молекул дцРНК.
Фигура 37. Пониженная продуктивность тли после кормления искусственным кормом с добавкой кпРНК для снижения экспрессии генов MpC002 или MpRack-1 у зеленой персиковой тли. Верхняя панель (A): среднее число нимф на количество взрослых тлей после десятидневного периода с 100 мкл 50 нг/мкл кпРНК. Нижняя панель (B): процент тлей, выживших в течение пятидневного курса после питания на 100 мкл, содержащих 200 нг/мкл кпРНК MpC002, MpRack-1 или контрольной ledGFP.
ОПИСАНИЕ СПИСКА ПОСЛЕДОВАТЕЛЬНОСТЕЙ
SEQ ID NO:1 - Рибонуклеотидная последовательность кпРНК GFP.
SEQ ID NO:2 - Рибонуклеотидная последовательность кпРНК GUS.
SEQ ID NO:3 - Рибонуклеотидная последовательность кпРНК FAD2.1 N. benthamiana.
SEQ ID NO:4 - Нуклеотидная последовательность, кодирующая кпРНК GFP.
SEQ ID NO:5 - Нуклеотидная последовательность, кодирующая кпРНК GUS.
SEQ ID NO:6 - Нуклеотидная последовательность, кодирующая кпРНК FAD2.1 N. benthamiana.
SEQ ID NO:7 - Нуклеотидная последовательность, кодирующая GFP.
SEQ ID NO:8 - Нуклеотидная последовательность, кодирующая GUS.
SEQ ID NO:9 - Нуклеотидная последовательность, кодирующая FAD2.1 N. benthamiana.
SEQ ID NO:10 - Нуклеотидная последовательность, используемая для получения смысловой области GUS для конструкций, кодирующих молекулы шпилечных РНК, направленно взаимодействующие с мРНК GUS.
SEQ ID NO:11 - Нуклеотидная последовательность, используемая для получения смысловой области GUS для конструкции, кодирующей молекулу шпилечной РНК hpGUS[G:U].
SEQ ID NO:12 - Нуклеотидная последовательность, используемая для получения смысловой области GUS для конструкций, кодирующих молекулу шпилечной РНК hpGUS[1:4].
SEQ ID NO:13 - Нуклеотидная последовательность, используемая для получения смысловой области GUS для конструкций, кодирующих молекулу шпилечной РНК hpGUS[2:10].
SEQ ID NO:14 - Нуклеотидная последовательность нуклеотидов 781-1020 кодирующей белок области гена GUS.
SEQ ID NO:15 - Рибонуклеотидная последовательность шпилечной структуры (включая ее петлю) РНК hpGUS[wt].
SEQ ID NO:16 - Рибонуклеотид шпилечной структуры (включая ее петлю) РНК hpGUS[G:U].
SEQ ID NO:17 - Рибонуклеотид шпилечной структуры (включая ее петлю) РНК hpGUS[1:4].
SEQ ID NO:18 - Рибонуклеотид шпилечной структуры (включая ее петлю) РНК hpGUS[2:10].
SEQ ID NO:19 - Нуклеотидная последовательность кДНК, соответствующей гену EIN2 A. thaliana, рег. номер NM_120406.
SEQ ID NO:20 - Нуклеотидная последовательность кДНК, соответствующей гену CHS A. thaliana, рег. номер NM_121396, 1703 нт.
SEQ ID NO:21 - Нуклеотидная последовательность фрагмента ДНК, включающего 200 нт смысловую последовательность из кДНК, соответствующей гену EIN2 A. thaliana, фланкированную сайтами рестрикции.
SEQ ID NO:22 - Нуклеотидная последовательность фрагмента ДНК, включающего 200 нт смысловую последовательность EIN2, как в SEQ ID NO:21, за исключением того, что 43 C были заменены Т, используемая при конструировании hpEIN2[G:U].
SEQ ID NO:23 - Нуклеотидная последовательность фрагмента ДНК, включающего 200 нт смысловую последовательность из кДНК, соответствующей гену CHS A. thaliana, фланкированную сайтами рестрикции.
SEQ ID NO:24 - Нуклеотидная последовательность фрагмента ДНК, включающего 200 нт смысловую последовательность CHS, как в SEQ ID NO:23, за исключением того, что 65 C были заменены Т, используемая при конструировании hpCHS[G:U].
SEQ ID NO:25 - Нуклеотидная последовательность фрагмента ДНК, включающего 200 нт антисмысловую последовательность EIN2, в которой 50 C были заменены Т, используемая при конструировании hpEIN2[G:U/U:G].
SEQ ID NO:26 - Нуклеотидная последовательность фрагмента ДНК, включающего 200 нт антисмысловую последовательность CHS, в которой 49 C были заменены Т, используемая при конструировании hpCHS[G:U/U:G].
SEQ ID NO:27 - Нуклеотидная последовательность нуклеотидов 601-900 из кДНК, соответствующей гену EIN2 из A. thaliana (рег. номер NM_120406).
SEQ ID NO:28 - Нуклеотидная последовательность нуклеотидов 813-1112 из кДНК, соответствующей гену CHS от A. thaliana (рег. номер NM_121396).
SEQ ID NO:29 - Нуклеотидная последовательность комплемента нуклеотидов 652-891 из кДНК, соответствующей гену EIN2 из A. thaliana (рег. номер NM_120406).
SEQ ID NO:30 - Нуклеотидная последовательность комплемента нуклеотидов 804-1103 из кДНК, соответствующей гену CHS из A. thaliana.
SEQ ID NO:31 - Кодирующая белок FANCM I область кДНК Arabidopsis thaliana, рег. номер NM_001333162. Нуклеотиды 675-1174 из области-мишени (500 нуклеотидов).
SEQ ID NO:32 - Кодирующая белок FANCM I область кДНК Brassica napus. Нуклеотиды 896-1395 из области-мишени (500 пн).
SEQ ID NO:33 - Нуклеотидная последовательность, кодирующая hpFANCM-At[wt], направленно взаимодействующую с кодирующей белок FANCM I областью A. thaliana. Смысловая последовательность FANCM, нуклеотиды 38-537; последовательность петли, нуклеотиды 538-1306; антисмысловая последовательность FANCM, нуклеотиды 1307-1806.
SEQ ID NO:34 - Нуклеотидная последовательность, кодирующая hpFANCM-At[G:U], направленно взаимодействующую с кодирующей белок FANCM I областью A. thaliana. Смысловая последовательность FANCM, нуклеотиды 38-537; последовательность петли, нуклеотиды 538-1306; антисмысловая последовательность FANCM, нуклеотиды 1307-1806.
SEQ ID NO:35 - Нуклеотидная последовательность, кодирующая hpFANCM-Bn[wt], направленно взаимодействующую с кодирующей белок FANCM I областью B. napus. Смысловая последовательность FANCM, нуклеотиды 34-533; последовательность петли, нуклеотиды 534-1300; антисмысловая последовательность FANCM, нуклеотиды 1301-1800.
SEQ ID NO:36 - Нуклеотидная последовательность, кодирующая hpFANCM-Bn[G:U], направленно взаимодействующую с кодирующей белок FANCM I областью B. napus. Смысловая последовательность FANCM, нуклеотиды 34-533; последовательность петли, нуклеотиды 534-1300; антисмысловая последовательность FANCM, нуклеотиды 1301-1800.
SEQ ID NO:37 - Нуклеотидная последовательность кодирующей белок области кДНК, соответствующей гену DDM1 B. napus; рег. номер XR_001278527.
SEQ ID NO:38 - Нуклеотидная последовательность ДНК, кодирующей hpDDM1-Bn[wt], направленно взаимодействующую с кодирующей белок областью DDM1 B. napus.
SEQ ID NO:39 - Нуклеотидная последовательность, кодирующая hpDDM1-Bn[G:U], направленно взаимодействующую с кодирующей белок областью DDM1 B. napus. Смысловая последовательность DDM1, нуклеотиды 35-536; последовательность петли, нуклеотиды 537-1304; антисмысловая последовательность DDM1, нуклеотиды 1305-1805.
SEQ ID NO:40 - кДНК EGFP.
SEQ ID NO:41 - Нуклеотидная последовательность кодирующей области hpEGFP[wt], с порядком антисмысловая/петля/смысловая относительно промотора.
SEQ ID NO:42 - Нуклеотидная последовательность кодирующей области hpEGFP[G:U], которая содержит 157 замен C на T в смысловой последовательности EGFP.
SEQ ID NO:43 - Нуклеотидная последовательность кодирующей области ledEGFP[wt], которая не содержит замен C на T в смысловой последовательности EGFP.
SEQ ID NO:44 - Нуклеотидная последовательность кодирующей области ledEGFP[G:U], которая содержит 162 замены C на T в смысловой последовательности EGFP.
SEQ ID NO:45 - Нуклеотидная последовательность, используемая для получения смысловой области GUS для конструкции, кодирующей молекулу шпилечной РНК hpGUS[G:U] без фланкирующих сайтов рестрикции.
SEQ ID NO:46 - Нуклеотидная последовательность, используемая для получения смысловой области GUS для конструкций, кодирующих молекулу шпилечной РНК hpGUS[1:4] без фланкирующих сайтов рестрикции.
SEQ ID NO:47 - Нуклеотидная последовательность, используемая для получения смысловой области GUS для конструкций, кодирующих молекулу шпилечной РНК hpGUS[2:10] без фланкирующих сайтов рестрикции.
SEQ ID NO:48 - Нуклеотидная последовательность фрагмента ДНК, включающего 200 нт смысловую последовательность EIN2, как в SEQ ID NO:21, за исключением того, что 43 C были заменены Т, используемая при конструировании hpEIN2[G:U] без фланкирующих последовательностей.
SEQ ID NO:49 - Нуклеотидная последовательность фрагмента ДНК, включающего 200 нт смысловую последовательность CHS, как в SEQ ID NO:23, за исключением того, что 65 C были заменены Т, используемая при конструировании hpCHS[G:U] без фланкирующих последовательностей.
SEQ ID NO:50 - Нуклеотидная последовательность фрагмента ДНК, включающего 200 нт антисмысловую последовательность EIN2 с заменой 50 C на Т, используемая при конструировании hpEIN2[G:U/U:G] без фланкирующих последовательностей
SEQ ID NO:51 - Нуклеотидная последовательность фрагмента ДНК, включающего 200 нт антисмысловую последовательность CHS с заменой 49 C на Т, используемая при конструировании hpCHS[G:U/U:G] без фланкирующих последовательностей.
SEQ ID NO:52 - Олигонуклеотидный праймер, используемый для амплификации 200 пн смысловой последовательности GUS (GUS-WT-F)
SEQ ID NO:53 - Олигонуклеотидный праймер, используемый для амплификации 200 пн смысловой последовательности GUS (GUS-WT-R)
SEQ ID NO:54 - Олигонуклеотидный праймер (прямой), используемый для получения фрагмента hpGUS[G:U] с заменой всех C на T (GUS-GU-F)
SEQ ID NO:55 - Олигонуклеотидный праймер (обратный), используемый для получения фрагмента hpGUS[G:U] с заменой всех C на T (GUS-GU-R)
SEQ ID NO:56 - Олигонуклеотидный праймер (прямой), используемый для получения фрагмента hpGUS[1:4] с заменой каждого 4-го нуклеотида (GUS-4M-F)
SEQ ID NO:57 - Олигонуклеотидный праймер (обратный), используемый для получения фрагмента hpGUS[1:4] с заменой каждого 4-го нуклеотида (GUS-4M-R)
SEQ ID NO:58 - Олигонуклеотидный праймер (прямой), используемый для получения фрагмента hpGUS[2:10] с заменой каждого 9-го и 10-го нуклеотида (GUS-10M-F)
SEQ ID NO:59 - Олигонуклеотидный праймер (обратный), используемый для получения фрагмента hpGUS[2:10] с заменой каждого 9-го и 10-го нуклеотида (GUS-10M-R)
SEQ ID NO:60 - Нуклеотидная последовательность, кодирующая прямой праймер (35-F3)
SEQ ID NO:61 - Нуклеотидная последовательность, кодирующая обратный праймер (GUSwt-R2)
SEQ ID NO:62 - Нуклеотидная последовательность, кодирующая прямой праймер (GUSgu-R2)
SEQ ID NO:63 - Нуклеотидная последовательность, кодирующая обратный праймер (GUS4m-R2)
SEQ ID NO:64 - Нуклеотидная последовательность, кодирующая прямой праймер (35-F2)
SEQ ID NO:65 - Нуклеотидная последовательность, кодирующая обратный праймер (35-R1)
SEQ ID NO:66 - Олигонуклеотидный праймер, используемый для амплификации 200 пн смысловой последовательности EIN2 дикого типа (EIN2wt-F)
SEQ ID NO:67 - Олигонуклеотидный праймер, используемый для амплификации 200 пн смысловой последовательности EIN2 дикого типа (EIN2wt-R)
SEQ ID NO:68 - Олигонуклеотидный праймер, используемый для амплификации 200 пн смысловой последовательности CHS дикого типа (CHSwt-F)
SEQ ID NO:69 - Олигонуклеотидный праймер, используемый для амплификации 200 пн смысловой последовательности CHS дикого типа (CHSwt-R)
SEQ ID NO:70 - Олигонуклеотидный праймер (прямой), используемый для получения фрагмента hpEIN2 [G:U] с заменой каждого C на T (EIN2gu-F)
SEQ ID NO:71 - Олигонуклеотидный праймер (обратный), используемый для получения фрагмента hpEIN2 [G:U] с заменой каждого C на T (EIN2gu-R)
SEQ ID NO:72 - Олигонуклеотидный праймер (прямой), используемый для получения фрагмента hpCHS [G:U] с заменой каждого C на T (CHSgu-F)
SEQ ID NO:73 - Олигонуклеотидный праймер (обратный), используемый для получения фрагмента hpCHS [G:U] с заменой каждого C на T (CHSgu-R)
SEQ ID NO:74 - Олигонуклеотидный праймер (прямой), используемый для получения фрагмента hpEIN2 [G:U/U:G] с заменой каждого C на T (asEIN2gu-F)
SEQ ID NO:75 - Олигонуклеотидный праймер (обратный), используемый для получения фрагмента hpEIN2 [G:U/U:G] с заменой каждого C на T (asEIN2gu-R)
SEQ ID NO:76 - Олигонуклеотидный праймер (прямой), используемый для получения фрагмента hpCHS [G:U/U:G] с заменой каждого C на T (asCHSgu-F)
SEQ ID NO:77 - Олигонуклеотидный праймер (обратный), используемый для получения фрагмента hpCHS [G:U/U:G] с заменой каждого C на T (asCHSgu-R)
SEQ ID NO:78 - Нуклеотидная последовательность, кодирующая прямой праймер (CHS-200-F2)
SEQ ID NO:79 - Нуклеотидная последовательность, кодирующая обратный праймер (CHS-200-R2)
SEQ ID NO:80 - Нуклеотидная последовательность, кодирующая прямой праймер (Actin2-For)
SEQ ID NO:81 - Нуклеотидная последовательность, кодирующая обратный праймер (Actin2-Rev)
SEQ ID NO:82 - Нуклеотидная последовательность, кодирующая прямой праймер (Top-35S-F2)
SEQ ID NO:83 - Нуклеотидная последовательность, кодирующая обратный праймер (Top-35S-R2)
SEQ ID NO:84 - Нуклеотидная последовательность, кодирующая прямой праймер (Link-35S-F2)
SEQ ID NO:85 - Нуклеотидная последовательность, кодирующая обратный праймер (Link-EIN2-R2)
SEQ ID NO:86 - Рибонуклеотидная последовательность смыслового si22
SEQ ID NO:87 - Рибонуклеотидная последовательность антисмыслового si22
SEQ ID NO:88 - Рибонуклеотидная последовательность прямого праймера
SEQ ID NO:89 - Рибонуклеотидная последовательность обратного праймера
SEQ ID NO:90 - Рибонуклеотидная последовательность прямого праймера
SEQ ID NO:91 - Рибонуклеотидная последовательность обратного праймера
SEQ ID NO:92 - Возможные модификации молекул дцРНК
SEQ ID NO:93 - Нуклеотидная последовательность кДНК, соответствующей гену DDM1 Brassica napus (рег. номер XR_001278527).
SEQ ID NO:94 - Нуклеотидная последовательность химерной ДНК, кодирующей конструкцию шпилечной РНКи (шРНК), направленно воздействующую на ген DDM1 B. napus.
SEQ ID NO:95 - Нуклеотидная последовательность химерной ДНК, кодирующей конструкцию шпилечной РНКи (шРНК) с парами оснований G:U, направленно воздействующую на ген DDM1 B. napus.
SEQ ID NO:96 - Нуклеотидная последовательность химерной ДНК, кодирующей конструкцию кпРНК, направленно воздействующую на ген DDM1 B. napus.
SEQ ID NO:97 - Нуклеотидная последовательность кДНК, соответствующей гену FANCM A. thaliana (рег. номер NM_001333162).
SEQ ID NO:98 - Нуклеотидная последовательность химерной ДНК, кодирующей конструкцию шпилечной РНКи (шРНК), направленно воздействующую на ген FANCM A. thaliana.
SEQ ID NO:99 - Нуклеотидная последовательность химерной ДНК, кодирующей конструкцию шпилечной РНКи (шРНК) с парами оснований G:U, направленно воздействующую на ген FANCM A. thaliana.
SEQ ID NO:100 - Нуклеотидная последовательность химерной ДНК, кодирующей конструкцию кпРНК, направленно воздействующую на ген FANCM A. thaliana.
SEQ ID NO:101 - Нуклеотидная последовательность кДНК, соответствующей гену FANCM B. napus (рег. номер XM_022719486.1).
SEQ ID NO:102 - Нуклеотидная последовательность химерной ДНК, кодирующей конструкцию шпилечной РНКи (шРНК), направленно воздействующую на ген FANCM B. napus.
SEQ ID NO:103 - Нуклеотидная последовательность химерной ДНК, кодирующей конструкцию шпилечной РНКи (шРНК) с парами оснований G:U, направленно воздействующую на ген FANCM B. napus.
SEQ ID NO:104 - Нуклеотидная последовательность химерной ДНК, кодирующей конструкцию кпРНК, направленно воздействующую на ген FANCM B. napus.
SEQ ID NO:105 - Нуклеотидная последовательность кодирующей белок области кДНК, соответствующей гену TOR Nicotiana benthamiana.
SEQ ID NO:106 - Нуклеотидная последовательность химерной ДНК, кодирующей конструкцию кпРНК, направленно воздействующую на ген TOR N. benthamiana.
SEQ ID NO:107 - Нуклеотидная последовательность кодирующей белок области кДНК, соответствующей гену ацетолактатсинтазы (ALS) ячменя, Hordeum vulgare (рег. номер LT601589).
SEQ ID NO:108 - Нуклеотидная последовательность химерной ДНК, кодирующей кпРНК, направленно воздействующую на ген ALS ячменя (H. vulgare).
SEQ ID NO:109 - Нуклеотидная последовательность кодирующей белок области кДНК, соответствующей гену HvNCED1 ячменя Hordeum vulgare (рег. номер AK361999).
SEQ ID NO:110 - Нуклеотидная последовательность кодирующей белок области кДНК, соответствующей гену HvNCED2 ячменя Hordeum vulgare (рег. номер DQ145931).
SEQ ID NO:111 - Нуклеотидная последовательность химерной ДНК, кодирующей конструкцию кпРНК, направленно воздействующую на гены NCED1 ячменя Hordeum vulgare и пшеницы Triticum aestivum.
SEQ ID NO:112 - нуклеотидная последовательность химерной ДНК, кодирующей конструкцию кпРНК, направленно воздействующую на гены NCED2 ячменя Hordeum vulgare и пшеницы Triticum aestivum.
SEQ ID NO:113 - Нуклеотидная последовательность кодирующей белок области кДНК, соответствующей гену ячменя, кодирующему ABA-OH-2 (Рег. номер DQ145933).
SEQ ID NO:114 - Нуклеотидная последовательность химерной ДНК, кодирующей конструкцию кпРНК, направленно воздействующую на гены ABA-OH-2 ячменя Hordeum vulgare и пшеницы Triticum aestivum.
SEQ ID NO:115 - Нуклеотидная последовательность кодирующей белок области кДНК, соответствующей гену A. thaliana, кодирующему EIN2 (At5g03280).
SEQ ID NO:116 - Нуклеотидная последовательность химерной ДНК, кодирующей конструкцию кпРНК, направленно воздействующую на ген EIN2 A. thaliana.
SEQ ID NO:117 - Нуклеотидная последовательность кодирующей белок области кДНК, соответствующей гену A. thaliana, кодирующему CHS (рег. номер NM_121396).
SEQ ID NO:118 - Нуклеотидная последовательность химерной ДНК, кодирующей конструкцию кпРНК, направленно воздействующую на ген CHS A. thaliana.
SEQ ID NO:119 - Нуклеотидная последовательность кодирующей белок области кДНК, соответствующей гену N-like L. angustifolius (Рег. номер XM_019604347).
SEQ ID NO:120 - Нуклеотидная последовательность химерной ДНК, кодирующей конструкцию кпРНК, направленно воздействующую на ген N-like L. angustifolius.
SEQ ID NO:121 - Нуклеотидная последовательность кодирующей белок области кДНК, соответствующей гену MLO Vitis pseudoreticulata (Рег. номер KR362912).
SEQ ID NO:122 - Нуклеотидная последовательность химерной ДНК, кодирующей первую конструкцию кпРНК, направленно воздействующую ген MLO Vitis.
SEQ ID NO:123 - Нуклеотидная последовательность кодирующей белок области кДНК, соответствующей гену MpC002 Myzus persicae.
SEQ ID NO:124 - Нуклеотидная последовательность кодирующей белок области кДНК, соответствующей гену MpRack-1 Myzus persicae.
SEQ ID NO:125 - Нуклеотидная последовательность химерной конструкции, кодирующей кпРНК, направленно воздействующую на ген C002 M.persicae.
SEQ ID NO:126 - Нуклеотидная последовательность химерной конструкции, кодирующей кпРНК, направленно воздействующую на ген Rack-1 M.persicae.
SEQ ID NO:127 - Нуклеотидная последовательность кДНК, соответствующей гену ABCwhite Helicoverpa armigera.
SEQ ID NO:128 - Нуклеотидная последовательность химерной ДНК, кодирующей конструкцию кпРНК, направленно воздействующую на ген ABC-переносчика white Helicoverpa armigera.
SEQ ID NO:129 - Нуклеотидная последовательность кДНК, соответствующей гену, подобному нейропептидам PBAN-типа Linepithema humile (XM_012368710).
SEQ ID NO:130 - Нуклеотидная последовательность химерной ДНК, кодирующей конструкцию кпРНК, направленно воздействующую на ген PBAN у аргентинских муравьев (рег. номер XM_012368710).
SEQ ID NO:131 - Нуклеотидная последовательность химерной ДНК, кодирующей конструкцию кпРНК, направленно воздействующую на ген, кодирующий каталитическую субъединицу A протонной АТФ-азы V-типа (рег. номер XM_023443547) L. cuprina.
SEQ ID NO:132 - Нуклеотидная последовательность химерной ДНК, кодирующей конструкцию кпРНК, направленно воздействующую на ген, кодирующий РНКазу 1/2 L. cuprina.
SEQ ID NO:133 - Нуклеотидная последовательность химерной ДНК, кодирующей конструкцию кпРНК, направленно воздействующую на ген, кодирующий хитинсинтазу L. cuprina.
SEQ ID NO:134 - Нуклеотидная последовательность химерной ДНК, кодирующей конструкцию кпРНК, направленно воздействующую на ген, кодирующий рецептор экдизона (EcR) L. cuprina.
SEQ ID NO:135 - Нуклеотидная последовательность химерной ДНК, кодирующей конструкцию кпРНК, направленно воздействующую на ген, кодирующий гамма-тубулин 1/1-подобный L. cuprina.
SEQ ID NO:136 - Ген-мишень TaMlo (AF384144).
SEQ ID NO:137 - Нуклеотидная последовательность химерной ДНК, кодирующей конструкцию кпРНК, направленно воздействующую на ген, кодирующий TaMlo.
SEQ ID NO:138 - Нуклеотидная последовательность кодирующей белок области кДНК, соответствующей гену MLO Vitis pseudoreticulata (рег. номер KR362912).
SEQ ID NO:139 - Нуклеотидная последовательность химерной ДНК, кодирующей первую конструкцию кпРНК, направленно воздействующую на ген MLO Vitis.
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Общие методы и определения
Если прямо не определено иное, все технические и научные термины, используемые в настоящем документе, должны иметь то же значение, которое обычно известно специалистам в данной области (например, в области культур клеток, молекулярной генетики, сайленсинга генов, химии белков и биохимии).
Если не указано иное, методы получения рекомбинантных белков, культур клеток и иммунологические методы, используемые в настоящем изобретении, являются стандартными методиками, хорошо известными специалистам в данной области. Такие методы описаны и объяснены в литературе в таких источниках, как J. Perbal, A Practical Guide to Molecular Cloning, John Wiley and Sons (1984), J. Sambrook et al., Molecular Cloning: A Laboratory Manual, Cold Spring Harbour Laboratory Press (1989), T.A. Brown (редактор), Essential Molecular Biology: A Practical Approach, Volumes 1 and 2, IRL Press (1991), D.M. Glover and B.D. Hames (редакторы), DNA Cloning: A Practical Approach, Volumes 1-4, IRL Press (1995 and 1996), and F.M. Ausubel et al. (редакторы), Current Protocols in Molecular Biology, Greene Pub. Associates and Wiley-Interscience (1988, включая все редакции по настоящее время), Ed Harlow and David Lane (редакторы) Antibodies: A Laboratory Manual, Cold Spring Harbour Laboratory, (1988), и J.E. Coligan et al. (редакторы) Current Protocols in Immunology, John Wiley & Sons (включая все редакции по настоящее время).
Термин "антисмысловой регуляторный элемент" или "антисмысловая последовательность рибонуклеиновой кислоты" или "антисмысловая последовательность РНК" при использовании в настоящем документе означает последовательность РНК, которая, по меньшей мере частично, комплементарна, по меньшей мере, части молекулы РНК-мишени, с которой она гибридизуется. В некоторых вариантах осуществления антисмысловая последовательность РНК модулирует (повышает или снижает) экспрессию или количество молекулы РНК-мишени, или ее активность, например, посредством снижения трансляции молекулы РНК-мишени. В некоторых вариантах осуществления антисмысловая последовательность РНК изменяет сплайсинг пре-мРНК-мишени, что приводит к другому варианту сплайсинга. Примеры компонентов антисмысловых последовательностей включают, без ограничения, олигонуклеотиды, олигонуклеозиды, аналоги олигонуклеотидов, миметики олигонуклеотидов и их химерные комбинации.
Термин "антисмысловая активность" используется в рамках настоящего описания для обозначения любой поддающейся обнаружению и/или измерению активности, связанной с гибридизацией антисмысловой последовательности РНК с ее молекулой РНК-мишени. Такое обнаружение и/или измерение может быть прямым или косвенным. В примере антисмысловую активность оценивают путем обнаружения и/или измерения количества транскрипта молекулы РНК-мишени. Антисмысловая активность может быть также обнаружена как изменение фенотипа, связанного с молекулой РНК-мишени. При использовании в настоящем документе термин "молекула РНК-мишени" относится к транскрипту гена, который подвергается модуляции антисмысловой последовательностью РНК согласно настоящему изобретению. Таким образом, "молекула РНК-мишени" может быть любой молекулой РНК, экспрессию или активность которой можно модулировать антисмысловой последовательностью РНК. Примеры молекул РНК-мишеней включают, без ограничения, РНК (в том числе, но без ограничения, пре-мРНК и мРНК или их части), транскрибируемую с ДНК, кодирующей целевой белок, рРНК, тРНК, малую ядерную РНК и миРНК, включая их формы-предшественники. РНК-мишень может быть геномной РНК патогена или вредителя, такого как вирус, или молекулой РНК, полученной из нее, такой как репликативная форма вирусного патогена, или их транскриптом. Например, молекула РНК-мишени может быть РНК из эндогенного гена (или мРНК, транскрибируемой с гена) или гена, который введен или может быть введен в эукариотическую клетку, экспрессия которой связана с конкретным фенотипом, признаком, нарушением или патологическим состоянием, или молекулой нуклеиновой кислоты из возбудителя инфекции. В примере молекула РНК-мишени находится в эукариотической клетке. В другом примере молекула РНК-мишени кодирует белок. В данном контексте антисмысловая активность может быть оценена путем обнаружения и или измерения количества белка-мишени, например, по его активности, такой как ферментная активность, или функции, отличной от ферментной, или по фенотипу, связанному с его функцией. При использовании в настоящем документе термин "белок-мишень" относится к белку, модулируемому антисмысловой последовательностью РНК согласно настоящему изобретению.
В некоторых вариантах осуществления антисмысловую активность оценивают путем обнаружения и/или измерения количества молекул РНК-мишеней и/или расщепленных молекул РНК-мишеней и/или альтернативно сплайсированных молекул РНК-мишеней.
Антисмысловая активность может быть обнаружена или измерена с помощью различных методов. Например, антисмысловая активность может быть обнаружена или оценена путем сравнения активности в конкретном образце и сравнения активности с активностью в контрольном образце.
Термин "направленное взаимодействие" используется в рамках настоящего описания для обозначения ассоциации антисмысловой последовательности РНК с конкретной молекулой РНК-мишени или конкретной областью нуклеотидов в молекуле РНК-мишени. В одном примере антисмысловая последовательность РНК согласно настоящему раскрытию обладает комплементарностью, по меньшей мере, с областью молекулы-мишени РНК. В данном контексте термин "комплементарность" относится к последовательности рибонуклеотидов, которая способна к спариванию оснований с последовательностью рибонуклеотидов на молекуле РНК-мишени посредством образования водородных связей между основаниями на рибонуклеотидах. Например, в РНК аденин (A) комплементарен урацилу (U), а гуанин (G) - цитозину (C).
В некоторых вариантах осуществления "комплементарное основание" относится к рибонуклеотиду антисмысловой последовательности РНК, который способен спариваться с рибонуклеотидом смысловой последовательности РНК в молекуле РНК согласно настоящему изобретению или в молекуле РНК-мишени. Например, если рибонуклеотид в определенном положении антисмысловой последовательности РНК способен образовывать водородную связь с рибонуклеотидом в определенном положении молекулы РНК-мишени, то положение водородной связи между последовательностью антисмысловой РНК и молекулой РНК-мишени считается комплементарным по этому рибонуклеотиду. Напротив, термин "некомплементарный" относится к паре рибонуклеотидов, которые не образуют водородные связи друг с другом или не поддерживают гибридизацию иным способом. Термин "комплементарный" также может использоваться для обозначения способности антисмысловой последовательности РНК гибридизоваться с другой нуклеиновой кислотой посредством комплементарности. В некоторых вариантах осуществления последовательность РНК и ее мишень комплементарны друг другу, если достаточное количество соответствующих положений в каждой молекуле занимают рибонуклеотиды, которые могут связываться друг с другом, обеспечивая стабильную ассоциацию между последовательностью антисмысловой РНК и последовательностью смысловой РНК в молекуле РНК согласно изобретению и/или в молекуле РНК-мишени. Специалисту в данной области известно, что включение некомплементарных нуклеотидов возможно без нарушения способности последовательности антисмысловой РНК и мишени оставаться в ассоциации. Таким образом, в настоящем документе описаны последовательности антисмысловой РНК, которые могут содержать приблизительно до 20% нуклеотидов, которые не спариваются друг с другом (то есть не являются комплементарными соответствующим нуклеотидам в мишени). Предпочтительно антисмысловые соединения содержат не больше чем приблизительно 15%, более предпочтительно не больше чем приблизительно 10%, наиболее предпочтительно не больше 5% некомплементарных нуклеотидов или не содержат их. Остальные рибонуклеотиды являются комплементарными или не нарушают иным образом гибридизацию (например, пары G:U или A:G) между последовательностью антисмысловой РНК и последовательностью смысловой РНК или молекулой РНК-мишени. Специалисту в данной области техники будет известно, что последовательности антисмысловых РНК, описанные в настоящем документе, по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% (полностью) комплементарны по меньшей мере с областью молекулы РНК-мишени.
При использовании в настоящем документе "химерная молекула РНК" относится к любой молекуле РНК, которая обычно не встречается в природе. В примере молекулы химерной РНК, раскрытые в настоящем документе, были модифицированы с созданием некомплементарностей в области(ях) дцРНК. Например, химерные молекулы РНК могут быть модифицированы с превращением цитозинов в урацилы. В одном примере химерные молекулы РНК были модифицированы путем обработки бисульфитом в течение некоторого времени и в условиях, достаточных для превращения неметилированных цитозинов в урацилы.
Специалисту в данной области будет известно, что различные комбинации рибонуклеотидов могут спариваться с образованием пары оснований. В настоящем изобретении рассматриваются как канонические, так и неканонические пары оснований. В одном примере спаривание оснований может включать A:T или G:C в молекуле ДНК или U:A или G:C в молекуле РНК. В другом примере спаривание оснований может включать A:G или G:T, или U:G.
Термин "каноническое спаривание оснований" при использовании в настоящем описании означает спаривание оснований между двумя нуклеотидами, которые представляют собой A:T или G:C в случае дезоксирибонуклеотидов или A:U или G:C в случае рибонуклеотидов.
Термин "неканоническое спаривание оснований" при использовании в настоящем описании означает взаимодействие между основаниями двух нуклеотидов, которое отличается от канонического спаривания оснований в контексте двух последовательностей ДНК или РНК. Например, неканоническое спаривание оснований включает спаривание между G и U (G:U) или между A и G (A:G). Примеры неканонического спаривания оснований включают пурин-пурин или пиримидин-пиримидин. Как правило, в рамках настоящего описания неканоническое спаривание оснований представляют собой G:U. Другими примерами неканонических пар азотистых оснований, менее предпочтительных, являются A:C, G:T, G:G и A:A.
Настоящее изобретение относится к РНК-компонентам, которые "гибридизуются" на протяжении ряда рибонуклеотидов. Специалистам в данной области будет известно, что такие термины, как "гибридизуется" и "гибридизующийся" используются для описания молекул, которые отжигаются на основе комплементарных последовательностей нуклеиновых кислот. Такие молекулы не должны быть на 100% комплементарными для гибридизации (то есть они не должны быть обязательно "полностью спаренными"). Например, при комплементарности последовательностей может присутствовать одно или более некомплементарных оснований. В одном примере компоненты РНК, определенные в настоящем документе, гибридизуются в жестких условиях гибридизации. Термин "жесткие условия гибридизации" относится к параметрам, с которыми знаком специалист в данной области, включая изменение температуры гибридизации в зависимости от длины молекулы РНК. Параметры гибридизации рибонуклеотидов можно найти в источниках, в которых описаны такие методы, Sambrook, et al. (выше), и Ausubel, et al. (выше). Например, жесткие условия гибридизации, используемые в настоящем документе, могут относиться к гибридизации при 65°C в гибридизационном буфере (3,5×SSC, 0,02% фиколла, 0,02% поливинилпирролидона, 0,02% бычьего сывороточного альбумина (BSA), 2,5 мМ NaH2PO4 (pH 7) (0,5% SDS, 2 мМ ЭДТА) с последующей одной или более промывками в 0,2×SSC, 0,01% BSA при 50°C. Более короткие компоненты РНК, такие как последовательности РНК длиной 20-24 нуклеотида, гибридизуются в условиях более низкой жесткости. Термин "условия гибридизации низкой жесткости" относится к параметрам, с которыми знаком специалист в данной области, включая изменение температуры гибридизации в зависимости от длины молекулы РНК. Например, условия гибридизации низкой жесткости, используемые в настоящем документе, могут относиться к гибридизации при 42°C в гибридизационном буфере (3,5×SSC, 0,02% фиколла, 0,02% поливинилпирролидона, 0,02% бычьего сывороточного альбумина (BSA), 2,5 мМ NaH2PO4 (pH 7), 0,5% SDS, 2 мМ ЭДТА) с последующей одной или более промывками в 0,2×SSC, 0,01% BSA при 30°C.
Настоящее изобретение также охватывает РНК-компоненты, которые "полностью спарены" на протяжении непрерывных рибонуклеотидов. Термин "полностью спаренный" используется в рамках настоящего описания для обозначения ряда непрерывных спариваний рибонуклеотидных оснований. Полностью спаренный ряд последовательных рибонуклеотидов не включает пропуски или неспаренные нуклеотиды. Термин "непрерывный" используется для обозначения ряда рибонуклеотидов. Рибонуклеотиды, включающие непрерывный ряд, соединяются непрерывным рядом фосфодиэфирных связей, причем каждый рибонуклеотид связан непосредственно со следующим.
Молекулы РНК настоящего изобретения включают смысловую последовательность и соответствующую антисмысловую последовательность. Родство между этими последовательностями определено в настоящем документе. Родство последовательностей и активность антисмысловой последовательности в отношении молекулы РНК-мишени также определены в настоящем документе.
Термин "ковалентно связанный" используется в рамках настоящего описания для обозначения связи между первыми и вторыми РНК-компонентами или любыми последовательностями РНК или рибонуклеотидами. Как будет известно специалисту в данной области, ковалентное соединение или связь являются химической связью, включающей распределение общих электронных пар между атомами. В примере первый и второй РНК-компоненты или смысловая последовательность РНК и антисмысловая последовательность РНК ковалентно связаны как часть одной цепи РНК, которая может складываться и гибридизоваться сама с собой посредством самокомплементарности. В этом примере компоненты ковалентно связаны на протяжении одного или более рибонуклеотидов фосфодиэфирными связями.
В рамках настоящего описания термин "гибридизация" означает спаривание комплементарных полинуклеотидов посредством спаривания комплементарных оснований. Хотя отсутствует ограничение конкретным механизмом, наиболее распространенный механизм спаривания включает образование водородных связей, которые могут образовываться по правилу Уотсона-Крика между комплементарными рибонуклеотидами.
При использовании в настоящем документе фраза "молекула РНК оказывает вредное воздействие на не относящийся к человеку организм" или подобные фразы означают, что молекула РНК-мишени молекулы РНК присутствует в не относящемся к человеку организме, и контакт клеток, экспрессирующих молекулу РНК, с молекулой РНК-мишени приводит к снижению уровней и/или активности молекулы РНК-мишени по сравнению с теми же клетками, не содержащими молекулу РНК. В варианте осуществления молекула РНК-мишени кодирует белок, важный для роста, воспроизводства или выживания. Как пример, если не относящийся к человеку организм является вредителем сельскохозяйственных культур или патогеном, или паразитом или патогеном животного, молекула РНК может оказывать вредное воздействие на питание вредителя или патогена, апоптоз клеток, дифференцировку клеток и развитие, продуктивность или потребность в половом размножении, формирование мышц, подергивание мышц, сокращение мышц, образование ювенильного гормона, регуляцию ювенильного гормона, регуляцию и транспорт ионов, поддержание потенциала клеточной мембраны, биосинтез аминокислот, деградацию аминокислот, сперматогенез, синтез феромонов, чувствительность к феромонам, образование антенн, формирование крыльев, формирование ног, формирование яиц, созревание личинок, образование пищеварительных ферментов, синтез гемолимфы, поддержание гемолимфы, передачу нервных импульсов, переход в личиночную стадию, окукливание, выход из окукливания, деление клеток, энергетический обмен, дыхание, метаболизм хитина, формирование структуры цитоскелета. В другом примере не относящийся к человеку организм представляет собой сорняк, и молекула РНК оказывает вредное воздействие на биосинтез аминокислот, фотосинтез, синтез жирных кислот, целостность клеточной мембраны, синтез пигментов или рост.
При использовании в настоящем документе фраза "молекула РНК оказывает благоприятное воздействие по меньшей мере на один симптом заболевания" или подобные фразы означают, что РНК-мишень молекулы присутствует у субъекта, и контакт клеток, экспрессирующих РНК-мишень, с молекулой РНК приводит к снижению уровня и/или активности РНК-мишени по сравнению с теми же клетками, в которых отсутствует молекула РНК. В одном варианте осуществления РНК-мишень кодирует белок, который играет роль при наличии заболевания. В одном варианте осуществления заболевание является раком или онкологическим заболеванием, инфекционным заболеванием, сердечно-сосудистым заболеванием, неврологическим заболеванием, прионным заболеванием, воспалительным заболеванием, аутоиммунным заболеванием, заболеванием легких, заболеванием почек, заболеванием печени, митохондриальным заболеванием, эндокринными заболеваниями, заболеваниями и состояниями, связанными с репродукцией, и любыми другими показаниями, которые могут реагировать на уровень экспрессируемого продукта гена в клетке или организме.
Молекулы РНК согласно настоящему изобретению и композиции, включающие их, могут вводить субъекту. Такие термины, как "субъект", "пациент" или "индивид" являются терминами, которые в некотором контексте могут использоваться попеременно в настоящем описании. В примере субъект является млекопитающим. Млекопитающее может быть домашним животным, таким как собака или кошка, или сельскохозяйственным животным, таким как лошадь или корова. В одном примере субъектом является человек. Например, субъект может быть взрослым. В другом примере субъект может быть ребенком. В другом примере субъект может быть подростком. В другом примере молекулы РНК согласно настоящему изобретению и композиции, включающих их, могут вводить насекомому. В другом примере молекулы РНК согласно настоящему изобретению и композиции, включающих их, могут вводить в растение. В другом примере молекулы РНК согласно настоящему изобретению и композиции, включающих их, могут вводить в клетке гриба или популяцию.
Термин "и/или", например, "X и/или Y", следует понимать как означающий "X и Y" или "X или Y", при этом он должен использоваться в рамках прямой поддержки обоих значений или одного из значений.
При использовании в настоящем документе термин приблизительно, если не указано обратное, относится к +/-20%, более предпочтительно +/-10% от указанного значения.
В тексте настоящего описания слово "включают" или его вариации, такие как "включает" или "включающий", предусматривают включение указанного элемента, целого числа или этапа или группы элементов, целых чисел или этапов, но не исключение какого-либо другого элемента, целого числа или этапа или группы элементов, целых чисел или этапов.
Молекула РНК
В некоторых вариантах осуществления молекулы РНК настоящего изобретения включают первый РНК-компонент, который ковалентно связан со вторым РНК-компонентом. В предпочтительных вариантах осуществления молекула РНК самогибридизуется или сворачивается с формированием "гантели" или структуры кпРНК, например, см. Фигуру 1. В варианте осуществления молекула дополнительно включает одно или более следующего:
- соединительная рибонуклеотидная последовательность, ковалентно связывающая первый и второй РНК-компоненты;
- 5'-лидерная последовательность; и,
- 3'-трейлерная последовательность.
В варианте осуществления первый РНК-компонент состоит, в порядке от 5' к 3', из первого 5'-рибонуклеотида, первой последовательности РНК и первого 3'-рибонуклеотида, где первые 5' и 3' рибонуклеотиды спариваются друг с другом в молекуле РНК, где первая последовательность РНК включает первую смысловую рибонуклеотидную последовательность по меньшей мере из 20 последовательных рибонуклеотидов, первую последовательность петли по меньшей мере из 4 рибонуклеотидов и первую антисмысловую рибонуклеотидную последовательность по меньшей мере из 20 последовательных рибонуклеотидов, где первая антисмысловая рибонуклеотидная последовательность гибридизуется с первой смысловой рибонуклеотидной последовательностью в молекуле РНК, где первая антисмысловая рибонуклеотидная последовательность способна гибридизоваться с первой областью молекулы РНК-мишени.
В другом варианте осуществления первый РНК-компонент состоит, в порядке от 5' к 3', из первого 5'-рибонуклеотида, первой последовательности РНК и первого 3'-рибонуклеотида, где первые 5' и 3' рибонуклеотиды спариваются друг с другом в молекуле РНК, где первая последовательность РНК включает первую смысловую рибонуклеотидную последовательность по меньшей мере из 20 последовательных рибонуклеотидов, первую последовательность петли по меньшей мере из 4 рибонуклеотидов и первую антисмысловую рибонуклеотидную последовательность по меньшей мере из 20 последовательных рибонуклеотидов, где первая антисмысловая рибонуклеотидная последовательность полностью спаривается с первой смысловой рибонуклеотидной последовательностью в молекуле РНК, где первая антисмысловая рибонуклеотидная последовательность идентична по последовательности комплементу первой области молекулы РНК-мишени. Пример такого первого РНК-компонента этих двух вариантов осуществления схематично показан на левой половине Фигуры 1A или правой половине Фигуры 1B.
В другом варианте осуществления первый РНК-компонент состоит из первого 5'-рибонуклеотида, первой последовательности РНК и первого 3'-рибонуклеотида, где первые 5' и 3' рибонуклеотиды спариваются друг с другом в первом РНК-компоненте, где первая последовательность РНК включает первую смысловую рибонуклеотидную последовательность, первую последовательность петли по меньшей мере из 4 рибонуклеотидов и первую антисмысловую рибонуклеотидную последовательность, где первая смысловая рибонуклеотидная последовательность и первая антисмысловая рибонуклеотидная последовательность содержат по меньшей мере 20 последовательных рибонуклеотидов, при этом по меньшей мере 20 последовательных рибонуклеотидов первой смысловой рибонуклеотидной последовательности полностью спариваются по меньшей мере с 20 последовательными рибонуклеотидами первой антисмысловой рибонуклеотидной последовательности, где по меньшей мере 20 последовательных рибонуклеотидов первой смысловой рибонуклеотидной последовательности по существу идентичны по последовательности первой области молекулы РНК-мишени.
В этих вариантах осуществления пара оснований, образованная между первым 5'-рибонуклеотидом и первым 3'-рибонуклеотидом, считается концевой парой оснований области дцРНК, сформированной в результате самогибридизации первого РНК-компонента, т.е. он определяет конец области дцРНК.
В варианте осуществления первая смысловая последовательность обладает существенной идентичностью последовательности с областью РНК-мишени, где идентичность может быть с последовательностью длиной меньше 20 нуклеотидов. В варианте осуществления по меньшей мере 15, по меньшей мере 16, по меньшей мере 17, по меньшей мере 18 или по меньшей мере 19 последовательных рибонуклеотидов, предпочтительно по меньшей мере 20 последовательных рибонуклеотидов первой смысловой рибонуклеотидной последовательности и первой области молекулы РНК-мишени являются идентичными по последовательности по меньшей мере на 60%, по меньшей мере на 65%, по меньшей мере на 70%, по меньшей мере на 75%, по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или на 99%. В другом варианте осуществления, по меньшей мере 15, по меньшей мере 16, по меньшей мере 17, по меньшей мере 18, по меньшей мере 19 последовательных рибонуклеотидов первой смысловой рибонуклеотидной последовательности и первой области молекулы РНК-мишени идентичны на 100%. В варианте осуществления, первые 3, первые 4, первые 5, первые 6 или первые 7 рибонуклеотидов с 5'-конца первой смысловой рибонуклеотидной последовательности на 100% идентичны области молекулы РНК-мишени, а остальные рибонуклеотиды являются идентичными молекуле РНК-мишени по меньшей мере на 60%, по меньшей мере на 65%, по меньшей мере на 70%, по меньшей мере на 75%, по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99%.
В варианте осуществления по меньшей мере 20 последовательных рибонуклеотидов первой смысловой рибонуклеотидной последовательности и первой области молекулы РНК-мишени являются идентичными по меньшей мере на 60%, по меньшей мере на 65%, по меньшей мере на 70%, по меньшей мере на 75%, по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99%. Опять же, в этом варианте осуществления первые 3, первые 4, первые 5, первые 6 или первые 7 рибонуклеотидов могут быть на 100% идентичными области молекулы РНК-мишени, а остальные рибонуклеотиды являются идентичными молекуле РНК-мишени по меньшей мере на 60%, по меньшей мере на 65%, по меньшей мере на 70%, по меньшей мере на 75%, по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99%. В другом варианте осуществления по меньшей мере 20 последовательных рибонуклеотидов первой смысловой рибонуклеотидной последовательности и первой области молекулы РНК-мишени идентичны на 100%.
В варианте осуществления первая антисмысловая последовательность обладает существенной идентичностью последовательности с комплементом области РНК-мишени, где идентичность может быть с последовательностью, содержащей меньше 20 нуклеотидов по длине комплемента. В варианте осуществления по меньшей мере 15, по меньшей мере 16, по меньшей мере 17, по меньшей мере 18 или по меньшей мере 19 последовательных рибонуклеотидов, предпочтительно по меньшей мере 20 последовательных рибонуклеотидов первой антисмысловой рибонуклеотидной последовательности и комплемент первой области молекулы РНК-мишени являются идентичными по последовательности по меньшей мере на 60%, по меньшей мере на 65%, по меньшей мере на 70%, по меньшей мере на 75%, по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или на 99%. В другом варианте осуществления, по меньшей мере 15, по меньшей мере 16, по меньшей мере 17, по меньшей мере 18, по меньшей мере 19 последовательных рибонуклеотидов первой антисмысловой рибонуклеотидной последовательности и комплемент первой области молекулы РНК-мишени идентичны на 100%. В варианте осуществления первые 3, первые 4, первые 5, первые 6 или первые 7 рибонуклеотидов с 5'-конца первой антисмысловой рибонуклеотидной последовательности на 100% идентичны комплементу области молекулы РНК-мишени, а остальные рибонуклеотиды по меньшей мере на 60%, по меньшей мере на 65%, по меньшей мере на 70%, по меньшей мере на 75%, по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% идентичны комплементу молекулы РНК-мишени.
В варианте осуществления по меньшей мере 20 последовательных рибонуклеотидов первой антисмысловой рибонуклеотидной последовательности и комплемент первой области молекулы РНК-мишени являются идентичными по меньшей мере на 60%, по меньшей мере на 65%, по меньшей мере на 70%, по меньшей мере на 75%, по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99%. Опять же, в этом варианте осуществления первые 3, первые 4, первые 5, первые 6 или первые 7 рибонуклеотидов на 100% идентичны комплементу области молекулы РНК-мишени, а остальные рибонуклеотиды идентичны комплементу молекулы РНК-мишени по меньшей мере на 60%, по меньшей мере на 65%, по меньшей мере на 70%, по меньшей мере на 75%, по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99%. В другом варианте осуществления по меньшей мере 20 последовательных рибонуклеотидов первой антисмысловой рибонуклеотидной последовательности и первой области молекулы РНК-мишени идентичны на 100%.
В другом варианте осуществления второй РНК-компонент состоит, в порядке от 5' к 3', из второго 5'-рибонуклеотида, второй последовательности РНК и второго 3'-рибонуклеотида, где вторые 5' и 3' рибонуклеотиды спариваются, где вторая последовательность РНК включает вторую смысловую рибонуклеотидную последовательность, вторую последовательность петли по меньшей мере из 4 рибонуклеотидов и вторую антисмысловую рибонуклеотидную последовательность, где вторая смысловая рибонуклеотидная последовательность спаривается со второй антисмысловой рибонуклеотидной последовательностью. В этом варианте осуществления пара оснований, сформированная между вторым 5'-рибонуклеотидом и вторым 3'-рибонуклеотидом, считается концевой парой оснований области дцРНК, сформированной в результате самогибридизации второго РНК-компонента.
В варианте осуществления молекула РНК включает 5'-лидерную последовательность или 5'-удлиняющую последовательность, которые могут возникать в результате транскрипции с промотора в генетической конструкции с сайта начала транскрипции до начала полинуклеотида, кодирующего остальную часть молекулы РНК. Предпочтительно, что эта 5'-лидерная последовательность или 5'-удлиняющая последовательность относительно короткие по сравнению с остальной частью молекулы и могут быть удалены из молекулы РНК после транскрипции в варианте осуществления при обработке РНКазой. 5'-лидерная последовательность или 5'-удлиняющая последовательность могут быть в основном неспаренными или могут содержать одну или более структур стебель-петли. В этом варианте осуществления 5'-лидерная последовательность может состоять из последовательности рибонуклеотидов, которая ковалентно связана с первым 5'-рибонуклеотидом, если второй РНК-компонент связан с первым 3'-рибонуклеотидом, или со вторым 5'-рибонуклеотидом, если второй РНК-компонент связан с первым 5'-рибонуклеотидом. В варианте осуществления 5'-лидерная последовательность имеет длину по меньшей мере 10, по меньшей мере 20, по меньшей мере 30, по меньшей мере 100, по меньшей мере 200 рибонуклеотидов, предпочтительно до максимальной длины 250 рибонуклеотидов. В другом варианте осуществления 5'-лидерная последовательность имеет длину по меньшей мере 50 рибонуклеотидов. В варианте осуществления 5'-лидерная последовательность может действовать как удлиняющая последовательность для амплификации молекулы РНК путем подходящей реакции амплификации. В качестве варианта осуществления удлиняющая последовательность может облегчать амплификацию посредством полимеразы.
В другом варианте осуществления молекула РНК включает 3'-трейлерную последовательность или 3'-удлиняющую последовательность, которые могут возникать в результате транскрипции, продолжающейся до терминации транскрипции или сигнала полиаденилирования в конструкции, кодирующей молекулу РНК. 3'-трейлерная последовательность или 3'-удлиняющая последовательность могут включать polyA-хвост. Предпочтительно такая 3'-трейлерная последовательность или 3'-удлиняющая последовательность относительно короткие по сравнению с остальной частью молекулы и могут быть удалены из молекулы РНК после транскрипции в качестве варианта осуществления при обработке РНКазой. 3'-трейлерная последовательность или 3'-удлиняющая последовательность могут быть в основном неспаренными или могут содержать одну или более структур стебель-петли. В этом варианте осуществления 3'-трейлерная последовательность может состоять из последовательности рибонуклеотидов, которая ковалентно связана со вторым 3'-рибонуклеотидом, если второй РНК-компонент связан с первым 3'-рибонуклеотидом, или с первым 3'-рибонуклеотидом, если второй РНК-компонент связан с первым 5'-рибонуклеотидом. В варианте осуществления 3'-лидерная последовательность имеет длину по меньшей мере 10, по меньшей мере 20, по меньшей мере 30, по меньшей мере 100, по меньшей мере 200 рибонуклеотидов, предпочтительно до максимальной длины 250 рибонуклеотидов. В другом варианте осуществления 3'-лидерная последовательность имеет длину по меньшей мере 50 рибонуклеотидов. В варианте осуществления 3'-трейлерная последовательность может действовать как удлиняющая последовательность для амплификации молекулы РНК посредством подходящей реакции амплификации. В качестве варианта осуществления удлиняющая последовательность может облегчать амплификацию посредством полимеразы.
В варианте осуществления все рибонуклеотиды за исключением двух ковалентно связаны с двумя другими нуклеотидами, т.е. молекула РНК состоит только из одной цепи РНК, которая содержит самокомплементарные области, и, таким образом, имеет только один 5'-концевой нуклеотид и один 3'-концевой нуклеотид. В другом варианте осуществления все рибонуклеотиды кроме четырех ковалентно связаны с двумя другими нуклеотидами, то есть молекула РНК состоит из двух цепей РНК, которые содержат комплементарные области, которые гибридизуются, и, таким образом, имеет только два 5'-концевых нуклеотида и два 3'-концевых нуклеотида. В другом варианте осуществления каждый рибонуклеотид ковалентно связан с двумя другими нуклеотидами, то есть молекула РНК является кольцевой, а также имеет самокомплементарные области и поэтому не имеет 5'-концевого нуклеотида и 3'-концевого нуклеотида.
В одном варианте осуществления двухцепочечная область молекулы РНК может содержать одно или более выпетливаний, полученных из неспаренных нуклеотидов в смысловой последовательности РНК или антисмысловой последовательности РНК, или в обеих. В одном варианте осуществления молекула РНК содержит ряд выпетливаний. В качестве варианта осуществления двухцепочечная область молекулы РНК может содержать 2, 3, 4, 5, 6, 7, 8, 9, 10 или более выпетливаний. Каждое выпетливание может независимо содержать от одного, двух или более неспаренных нуклеотидов, до 10 нуклеотидов. Более длинные последовательности могут образовывать выпетливание из смысловых или антисмысловых последовательностей в области дцРНК, которые могут спариваться внутри или оставаться неспаренными. В другом варианте осуществления двухцепочечная область молекулы РНК не содержит выпетливаний, т.е. она полностью спарена по всей длине области дцРНК.
В другом варианте осуществления первая смысловая рибонуклеотидная последовательность ковалентно связана с первым 5'-рибонуклеотидом без каких-либо промежуточных нуклеотидов и/или первая антисмысловая рибонуклеотидная последовательность ковалентно связана с первым 3'-рибонуклеотидом без каких-либо промежуточных нуклеотидов. В другом варианте осуществления присутствует по меньшей мере один, по меньшей мере два, по меньшей мере три, по меньшей мере четыре, по меньшей мере пять, по меньшей мере шесть, по меньшей мере семь, по меньшей мере восемь, по меньшей мере девять, по меньшей мере 10 промежуточных нуклеотидов. Следует понимать, что такие промежуточные нуклеотиды не являются родственными по последовательности с молекулой РНК-мишени, но могут способствовать стабилизации спаривания оснований смежных смысловых и антисмысловых последовательностей.
В другом варианте осуществления 20 последовательных нуклеотидов первой смысловой рибонуклеотидной последовательности ковалентно связаны с первым 5'-рибонуклеотидом без каких-либо промежуточных нуклеотидов, и 20 последовательных нуклеотидов первой антисмысловой рибонуклеотидной последовательности ковалентно связаны с первым 3'-рибонуклеотидом без каких-либо промежуточных нуклеотидов. В другом варианте осуществления присутствует по меньшей мере один, по меньшей мере два, по меньшей мере три, по меньшей мере четыре, по меньшей мере пять, по меньшей мере шесть, по меньшей мере семь, по меньшей мере восемь, по меньшей мере девять, по меньшей мере 10 промежуточных нуклеотидов. Промежуточные нуклеотиды могут быть спарены в качестве части двухцепочечной области молекулы РНК, но не являются родственными по последовательности с РНК-мишенью. Они могут способствовать повышению стабильности двухцепочечной области или удерживать два конца молекулы РНК и не оставить неспаренными 5' и/или 3' конец.
В варианте осуществления вышеуказанные первый и второй РНК-компоненты включают соединительную рибонуклеотидную последовательность. В варианте осуществления соединительная рибонуклеотидная последовательность действует как спейсер между первой смысловой рибонуклеотидной последовательностью, которая по существу идентична по последовательности первой области молекулы РНК-мишени, и другими компонентами молекулы. Например, соединительная рибонуклеотидная последовательность может действовать как спейсер между этой областью и петлей. В другом варианте осуществления молекула РНК включает множество смысловых рибонуклеотидных последовательностей, которые по существу идентичны по последовательности первой области молекулы РНК-мишени и соединительной рибонуклеотидной последовательности, действующей как спейсер между этими последовательностями. В варианте осуществления по меньшей мере две, по меньшей мере три, по меньшей мере четыре, по меньшей мере пять, по меньшей мере шесть, по меньшей мере семь, по меньшей мере восемь, по меньшей мере девять, по меньшей мере 10 рибонуклеотидных последовательностей, которые по существу идентичны по последовательности первой области молекулы РНК-мишени, присутствуют в молекуле РНК, причем каждая отделена от другой(их) соединительной рибонуклеотидной последовательностью.
В варианте осуществления вышеуказанные молекулы РНК включают 5'-лидерную последовательность. В варианте осуществления 5'-лидерная последовательность состоит из последовательности рибонуклеотидов, которая ковалентно связана с первым 5'-рибонуклеотидом, если второй РНК-компонент связан с первым 3'-рибонуклеотидом, или со вторым 5'-рибонуклеотидом, если второй РНК-компонент связан с первым 5'-рибонуклеотидом. В варианте осуществления молекула РНК имеет модифицированный 5' или 3' конец в качестве варианта осуществления путем присоединения липидной группы, такой как холестерин или витамин, такой как биотин или полипептид. Такие модификации могут способствовать захвату молекулы РНК в эукариотическую клетку, где должна функционировать РНК.
В варианте осуществления соединительная рибонуклеотидная последовательность имеет длину меньше 100 рибонуклеотидов. В варианте осуществления соединительная рибонуклеотидная последовательность имеет длину меньше 50 рибонуклеотидов. В варианте осуществления соединительная рибонуклеотидная последовательность имеет длину меньше 20 рибонуклеотидов. В варианте осуществления соединительная рибонуклеотидная последовательность имеет длину меньше 10 рибонуклеотидов. В варианте осуществления соединительная рибонуклеотидная последовательность имеет длину меньше 5 рибонуклеотидов. В варианте осуществления соединительная рибонуклеотидная последовательность имеет длину от 1 до 100 рибонуклеотидов. В варианте осуществления соединительная рибонуклеотидная последовательность имеет длину от 1 до 50 рибонуклеотидов. В варианте осуществления соединительная рибонуклеотидная последовательность имеет длину от 1 до 20 рибонуклеотидов. В варианте осуществления соединительная рибонуклеотидная последовательность имеет длину от 1 до 10 рибонуклеотидов. В варианте осуществления соединительная рибонуклеотидная последовательность имеет длину от 1 до 5 рибонуклеотидов. В варианте осуществления рибонуклеотиды соединительной рибонуклеотидной последовательности не спарены. В предпочтительном варианте осуществления все рибонуклеотиды соединительной рибонуклеотидной последовательности спарены или спарены все кроме 1, 2 или 3 рибонуклеотидов.
В варианте осуществления первый или второй РНК-компонент включает шпилечную структуру. В предпочтительном варианте осуществления и первый, и второй РНК-компоненты включают шпилечную структуру. В этих вариантах осуществления шпилечная структура может быть стебель-петлей. Таким образом, в варианте осуществления молекула РНК может включать первый и второй РНК-компоненты, каждый из которых включает шпилечную структуру, где шпильки ковалентно связаны линкерной последовательностью. См., например, Фигуру 1. В варианте осуществления линкерная последовательность представляет собой одну или более неспаренных рибонуклеиновых кислот. В варианте осуществления линкерная последовательность составляет от 1 до 10 неспаренных рибонуклеотидов.
В варианте осуществления молекула РНК имеет двойную шпилечную структуру, т.е. "структуру кпРНК" или "структуру гантели". В этом варианте осуществления первая шпилька является первым РНК-компонентом, а вторая шпилька является вторым РНК-компонентом. В этих вариантах осуществления ковалентно соединены либо первый 3'-рибонуклеотид и второй 5'-рибонуклеотид, либо второй 3'-рибонуклеотид и первый 5'-рибонуклеотид, но не все одновременно. В этом варианте осуществления другие 5'/3' рибонуклеотиды могут быть отделены ником (т.е. разрывом в молекуле дцРНК, в котором между 5'/3' рибонуклеотидами отсутствует фосфодиэфирная связь). Вариант осуществления такого типа конфигурации показан на Фигуре 1B. В другом варианте осуществления соответствующие 5'/3' рибонуклеотиды могут быть отделены петлей. Длиная 5'-лидерной и 3'-трейлерной последовательностей может быть одинаковой или разной. В качестве варианта осуществления 5'-лидер может быть приблизительно на 5, 10, 15, 20, 25, 50, 100, 200, 500 рибонуклеотидов длиннее, чем 3'-трейлерная последовательность, или наоборот.
В вариантах осуществления, где молекула РНК имеет двойную шпилечную структуру, вторая шпилька (в дополнение к первой шпилечной структуре) включает смысловую последовательность РНК и антисмысловую последовательность РНК, которые по существу идентичны по последовательности области молекулы РНК-мишени или ее комплемента, соответственно. В варианте осуществления каждая шпилька содержит ряд рибонуклеотидов, которые по существу идентичны по последовательности области той же молекулы РНК-мишени. В варианте осуществления каждая шпилька содержит ряд рибонуклеотидов, которые по существу идентичны по последовательности различным областям той же молекулы РНК-мишени. В варианте осуществления каждая шпилька содержит ряд рибонуклеотидов, которые по существу идентичны по последовательности области разных молекул РНК-мишеней, т.е. молекула РНК может применяться для снижения экспрессии и/или активности двух молекул РНК-мишеней, которые могут не иметь родственные последовательности.
В каждой шпильке двойной шпилечной структуры молекулы РНК порядок смысловых и антисмысловых последовательностей РНК в каждой шпильке, в порядке от 5' к 3', может независимо включать сначала смысловую, затем антисмысловую, или сначала антисмысловую, затем смысловую. В предпочтительных вариантах осуществления порядок смысловых и антисмысловых последовательностей в двойной шпилечной структуре молекулы РНК является либо антисмысловая-смысловая-смысловая-антисмысловая, где две смысловых последовательности являются смежными (Фигура 1A), или смысловая-антисмысловая-антисмысловая-смысловая, где две антисмысловых последовательности являются смежными (Фигура 1B).
В варианте осуществления молекула РНК может включать, в порядке от 5' к 3', 5'-лидерную последовательность, первую петлю, смысловую последовательность РНК, вторую петлю и 3'-трейлерную последовательность, где 5' и 3'-лидерные последовательности ковалентно связаны со смысловой цепью, с формированием последовательности дцРНК. В варианте осуществления 5'-лидерная и 3'-трейлерная последовательности не связаны ковалентно друг с другом. В варианте осуществления 5'-лидерная и 3'-трейлерная последовательности отделены ником. В варианте осуществления 5'-лидерная и 3'-трейлерная последовательности лигированы друг с другом, с получением молекулы РНК с замкнутой структурой. В другом варианте осуществления 5'-лидерная и 3'-трейлерная последовательности отделены петлей.
Термин "петля" используется в рамках настоящего описания для обозначения структуры петли в молекуле РНК, раскрытой в настоящем документе, которая сформирована рядом некомплементарных рибонуклеотидов. Петли обычно следуют за рядом пар оснований между первым и вторым РНК-компонентами или соединяют смысловую последовательность РНК и антисмысловую последовательность РНК в одном из первого и второго компонентов РНК или в обоих. В варианте осуществления все рибонуклеотиды петли не комплементарны, обычно в случае более коротких петель из 4-10 рибонуклеотидов. В других вариантах осуществления некоторые рибонуклеотиды в одной или более петель комплементарны и способны спариваться в последовательности петли, при условии, что эти спаривания позволяют формироваться структуре петли. Например, по меньшей мере 5%, по меньшей мере 10% или по меньшей мере 15% рибонуклеотидов петли комплементарны. Варианты осуществления петель включают стебель-петли или шпильки, псевдоузлы и тетрапетли.
В варианте осуществления молекула РНК включает только две петли. В другом варианте осуществления молекула РНК включает по меньшей мере две, по меньшей мере три, по меньшей мере 4, по меньшей мере 5, по меньшей мере 6, по меньшей мере 7, по меньшей мере 8, по меньшей мере 9 или по меньшей мере 10 петель, предпочтительно максимум до 10 петель. Например, молекула РНК может включать 4 петли.
В настоящем изобретении предусмотрены петли разных размеров. Например, петли могут включать 4, 5, 6, 7, 8, 9, 10, 11 или 12 рибонуклеотидов. В других вариантах осуществления петли включают 15, 20, 25 или 30 нуклеотидов. В варианте осуществления один или все последовательности петель имеют длину больше 20 нуклеотидов. В других вариантах осуществления петли больше, например, включают 50, 100, 150, 200 или 300 рибонуклеотидов. В варианте осуществления петли включают 160 рибонуклеотидов. В другом варианте осуществления, менее предпочтительном, петли включают 200, 500, 700 или 1000 рибонуклеотидов при условии, что петли не препятствуют гибридизации смысловой и антисмысловой последовательностей РНК. В варианте осуществления каждая из петель содержит одинаковое количество рибонуклеотидов. Например, петли могут иметь длину от 100 до 1000 рибонуклеотидов. Например, петли могут иметь длину от 600 до 1000 рибонуклеотидов. Например, петли могут иметь длину от 4 до 1000 рибонуклеотидов. Например, петли предпочтительно имеют длину от 4 до 50 рибонуклеотидов. В другом варианте осуществления петли включают разное количество рибонуклеотидов.
В другом варианте осуществления одна или более петель включают интрон, который может быть вырезан из молекулы РНК. В варианте осуществления интрон происходит из гена растения. Примеры интронов включают интрон 3 из алкогольдегидрогеназы кукурузы 1 (Adh1) (GenBank: AF044293), интрон 4 из альфа-субъединицы бета-конглицинина сои (GenBank: AB051865); один из интронов гена rbcS-3A малой субъединицы рибулозо-1,5-бисфосфаткарбоксилазы гороха (RBC) (GenBank: X04333). Другие варианты осуществления подходящих интронов обсуждаются в (McCullough and Schuler, 1997; Smith et al., 2000).
В различных вариантах осуществления петля может быть на конце по меньшей мере 3, по меньшей мере 4, по меньшей мере 5, по меньшей мере 6, по меньшей мере 7, по меньшей мере 8, по меньшей мере 9, по меньшей мере 10 последовательных пар оснований, которые могут быть каноническими парами оснований или могут включать одну или более неканонических пар оснований. В других вариантах осуществления, менее предпочтительных, особенно в случае клеток позвоночных животных, петля может быть на конце по меньшей мере 20, 30, 50, 100, 200, 500 или больше последовательных пар оснований.
В другом варианте осуществления молекула РНК включает две или более смысловых рибонуклеотидных последовательностей, при этом антисмысловые рибонуклеотидные последовательности, полностью спаренные с ними, каждая из которых является идентичной по последовательности области молекулы РНК-мишени. Например, молекула РНК может включать 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12 или больше смысловых рибонуклеотидных последовательностей, и антисмысловые рибонуклеотидные последовательности, полностью спаренные с ними, где каждая из смысловых рибонуклеотидных последовательностей независимо является идентичной по последовательности области молекулы РНК-мишени. В этом варианте осуществления любая одна или более, или все последовательности могут быть отделены соединительной рибонуклеотидной последовательностью(ями). В этом варианте осуществления любая одна или более, или все последовательности могут быть отделены петлей.
В варианте осуществления две или более смысловых рибонуклеотидных последовательностей идентичны по последовательности разным областям одной и той же молекулы РНК-мишени. Например, последовательности могут быть идентичными по меньшей мере 2, по меньшей мере 3, по меньшей мере 4, по меньшей мере 5, по меньшей мере 6 областям одной и той же молекулы-мишени. В другом варианте осуществления две или более смысловых рибонуклеотидных последовательностей идентичны по последовательности. В варианте осуществления две или больше смысловых рибонуклеотидных последовательностей идентичны по последовательности одной и той же области одной и той же молекулы РНК-мишени. В другом варианте осуществления две или более смысловых рибонуклеотидных последовательностей идентичны по последовательности разным молекулам РНК-мишеней. В качестве варианта осуществления последовательности могут быть идентичными по меньшей мере 2, по меньшей мере 3, по меньшей мере 4, по меньшей мере 5, по меньшей мере 6 областям разных молекул-мишеней.
В другом варианте осуществления две или более смысловых рибонуклеотидных последовательностей не имеют промежуточных (спейсерных) последовательностей петель.
В варианте осуществления молекула РНК имеет одну цепь рибонуклеотидов, имеющую 5'-конец, по меньшей мере одну смысловую рибонуклеотидную последовательность, которая имеет длину по меньшей мере 21 нуклеотид, антисмысловую рибонуклеотидную последовательность, которая полностью спарена с каждой смысловой рибонуклеотидной последовательностью на протяжении по меньшей мере 21 последовательного нуклеотида, по меньшей мере две последовательности петель и 3'-конец. В этом варианте осуществления рибонуклеотид на 5'-конце и рибонуклеотид на 3'-конце не связаны ковалентно напрямую, а скорее непосредственно примыкают к каждому спаренному.
В другом варианте осуществления последовательные пары оснований РНК-компонентов разделены по меньшей мере одним пропуском. В варианте осуществления "пропуск" обеспечивается неспаренным рибонуклеотидом. В другом варианте осуществления "пропуск" обеспечивается нелигированной 5'-лидерной последовательностью и/или 3'-трейлерной последовательностью. В этом варианте осуществления пропуск может именоваться "нелигированным пропуском". Некомплементарные основания и нелигированный пропуск(и) могут быть расположены в разных положениях молекулы РНК. В качестве варианта осуществления нелигированный пропуск может располагаться непосредственно после антисмысловой последовательности. В другом варианте осуществления нелигированный пропуск может располагаться вблизи от петли молекулы РНК. В другом варианте осуществления нелигированный пропуск расположен примерно равноудаленно по меньшей мере между двумя петлями.
В варианте осуществления молекула РНК получена из одной цепи РНК. В варианте осуществления одна цепь не является циклически замкнутой, например, она включает нелигированный пропуск. В другом варианте осуществления молекула РНК является циклически замкнутой молекулой. Замкнутые молекулы могут быть получены при лигировании вышеуказанной молекулы РНК, включающей нелигированный пропуск, например, с помощью РНК-лигазы.
В другом варианте осуществления молекула РНК включает 5'-и/или 3'-удлиняющую последовательность. Например, молекула РНК может включать 5'-удлиняющую последовательность, ковалентно связанную с первым 5'-рибонуклеотидом. В другом варианте осуществления молекула РНК включает 3'-удлиняющую последовательность, ковалентно связанную со вторым 3'-рибонуклеотидом. В другом варианте осуществления молекула РНК включает 5'-удлиняющую последовательность, ковалентно связанную с первым 5'-рибонуклеотидом, и 3'-удлиняющую последовательность, ковалентно связанную со вторым 3'-рибонуклеотидом.
В другом варианте осуществления молекула РНК включает 5'-удлиняющую последовательность, ковалентно связанную со вторым 5'-рибонуклеотидом. В другом варианте осуществления молекула РНК включает 3'-удлиняющую последовательность, ковалентно связанную с первым 3'-рибонуклеотидом. В другом варианте осуществления молекула РНК включает 5'-удлиняющую последовательность, ковалентно связанную со вторым 5'-рибонуклеотидом, и 3'-удлиняющую последовательность, ковалентно связанную с первым 3'-рибонуклеотидом.
В другом варианте осуществления молекула РНК может включать одно или более следующего:
- 5'-удлиняющую последовательность, ковалентно связанную с первым 5'-рибонуклеотидом;
- 3'-удлиняющую последовательность, ковалентно связанную со вторым 3'-рибонуклеотидом;
- 5'-удлиняющую последовательность, ковалентно связанную с первым 5'-рибонуклеотидом, и 3'-удлиняющую последовательность, ковалентно связанную со вторым 3'-рибонуклеотидом;
- 5'-удлиняющую последовательность, ковалентно связанную со вторым 5'-рибонуклеотидом;
- 3'-удлиняющую последовательность, ковалентно связанную с первым 3'-рибонуклеотидом;
- 5'-удлиняющую последовательность, ковалентно связанную со вторым 5'-рибонуклеотидом, и 3'-удлиняющую последовательность, ковалентно связанную с первым 3'-рибонуклеотидом.
Неканоническое спаривание оснований
В одном варианте осуществления молекулы РНК согласно настоящему изобретению содержат смысловую рибонуклеотидную последовательность и антисмысловую рибонуклеотидную последовательность, которые способны гибридизоваться друг с другом с образованием двухцепочечной области (дц)РНК с некоторым неканоническим спариванием оснований, то есть с комбинацией канонического и неканонического спаривания оснований. В одном варианте осуществления молекулы РНК согласно настоящему изобретению содержат две или более смысловых рибонуклеотидных последовательностей, каждая из которых способна гибридизоваться с областями одной (непрерывной) антисмысловой рибонуклеотидной последовательности с образованием области дцРНК с некоторым неканоническим спариванием оснований. См., например, Фигуру 1B. В одном варианте осуществления молекулы РНК согласно настоящему изобретению содержат две или более антисмысловых смысловых рибонуклеотидных последовательностей, каждая из которых способна гибридизоваться с областями одной (непрерывной) смысловой рибонуклеотидной последовательности с образованием области дцРНК с некоторым неканоническим спариванием оснований. См., например, Фигуру 1А. В одном варианте осуществления молекулы РНК согласно настоящему изобретению содержат две или более антисмысловых смысловых рибонуклеотидных последовательностей и две или более смысловых рибонуклеотидных последовательностей, где каждая антисмысловая рибонуклеотидная последовательность способна гибридизоваться с антисмысловой рибонуклеотидной последовательностью с образованием двух или более областей дцРНК, одна или обе из которых включают некоторое неканоническое спаривание оснований.
В следующих вариантах осуществления полноразмерная область дцРНК (т.е. полная область дцРНК) молекулы РНК согласно изобретению рассматривают как контекст для особенности, если присутствует только одна (непрерывная) область дцРНК, или для каждой из областей дцРНК молекулы РНК, если в молекуле РНК присутствуют две или более областей дцРНК. В варианте осуществления по меньшей мере 5% пар оснований в области дцРНК являются неканоническими парами оснований. В варианте осуществления по меньшей мере 6% пар оснований в области дцРНК являются неканоническими парами оснований. В варианте осуществления по меньшей мере 7% пар оснований в области дцРНК являются неканоническими парами оснований. В варианте осуществления по меньшей мере 8% пар оснований в области дцРНК являются неканоническими парами оснований. В варианте осуществления по меньшей мере 9% или 10% пар оснований в области дцРНК являются неканоническими парами оснований. В варианте осуществления по меньшей мере 11% или 12% пар оснований в области дцРНК являются неканоническими парами оснований. В варианте осуществления по меньшей мере 15% или приблизительно 15% пар оснований в области дцРНК являются неканоническими парами оснований. В варианте осуществления по меньшей мере 20% или приблизительно 20% пар оснований в области дцРНК являются неканоническими парами оснований. В варианте осуществления по меньшей мере 25% или приблизительно 25% пар оснований в области дцРНК являются неканоническими парами оснований. В варианте осуществления по меньшей мере 30% или приблизительно 30% пар оснований в области дцРНК являются неканоническими парами оснований. В каждом из этих вариантов осуществления предпочтительно, что максимум 40% пар оснований в области дцРНК являются неканоническими парами оснований, более предпочтительно максимум 35% пар оснований в области дцРНК являются неканоническими парами оснований, еще более предпочтительно максимум 30% пар оснований в области дцРНК являются неканоническими парами оснований. В варианте осуществления, менее предпочтительном, приблизительно 35% пар оснований в области дцРНК являются неканоническими парами оснований. В варианте осуществления, даже менее предпочтительном, приблизительно 40% пар оснований в области дцРНК являются неканоническими парами оснований. В каждом из вышеуказанных вариантов осуществления область дцРНК может включать или может не включать один или более неспаренных рибонуклеотидов в смысловой последовательности или в антисмысловой последовательности, или в обеих.
В варианте осуществления от 10% до 40% пар оснований в области дцРНК молекулы РНК согласно изобретению неканонические пары оснований. В варианте осуществления от 10% до 35% пар оснований в области дцРНК являются неканоническими парами оснований. В варианте осуществления от 10% до 30% пар оснований в области дцРНК являются неканоническими парами оснований. В варианте осуществления от 10% до 25% пар оснований в области дцРНК являются неканоническими парами оснований. В варианте осуществления от 10% до 20% пар оснований в области дцРНК являются неканоническими парами оснований. В варианте осуществления от 10% до 15% пар оснований в области дцРНК неканонические пары оснований. В варианте осуществления от 15% до 30% пар оснований в области дцРНК являются неканоническими парами оснований. В варианте осуществления от 15% до 25% пар оснований в области дцРНК являются неканоническими парами оснований. В варианте осуществления от 15% до 20% пар оснований в области дцРНК являются неканоническими парами оснований. В варианте осуществления от 5% до 30% пар оснований в области дцРНК являются неканоническими парами оснований. В варианте осуществления от 5% до 25% пар оснований в области дцРНК являются неканоническими парами оснований. В варианте осуществления от 5% до 20% пар оснований в области дцРНК являются неканоническими парами оснований. В варианте осуществления от 5% до 15% пар оснований в области дцРНК являются неканоническими парами оснований. В варианте осуществления от 5% до 10% пар оснований в области дцРНК являются неканоническими парами оснований. В каждом из вышеуказанных вариантов осуществления область дцРНК может включать или может не включать один или более неспаренных рибонуклеотидов в смысловой последовательности или в антисмысловой последовательности, или в обеих.
В варианте осуществления область дцРНК молекулы РНК согласно изобретению включает 20 последовательных пар оснований, где по меньшей мере одна пара оснований из 20 последовательных пар оснований является неканонической парой оснований. В варианте осуществления область дцРНК включает 20 последовательных пар оснований, где по меньшей мере 2 пары оснований из 20 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 20 последовательных пар оснований, где по меньшей мере 3 пары оснований из 20 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 20 последовательных пар оснований, где по меньшей мере 4 пары оснований из 20 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 20 последовательных пар оснований, где по меньшей мере 5 пар оснований из 20 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 20 последовательных пар оснований, где по меньшей мере 6 пар оснований из 20 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 20 последовательных пар оснований, где по меньшей мере 7 пар оснований из 20 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 20 последовательных пар оснований, где по меньшей мере 8 пар оснований из 20 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 20 последовательных пар оснований, где по меньшей мере 9 пар оснований из 20 последовательных пар оснований являются неканоническими парами оснований. В каждом из этих вариантов осуществления предпочтительно, что максимум 10 из 20 последовательных пар оснований в области дцРНК является неканоническими парами оснований, более предпочтительно максимум 9 из пар оснований в области дцРНК являются неканоническими парами оснований, еще более предпочтительно максимум 8 из пар оснований в области дцРНК являются неканоническими парами оснований, даже еще более предпочтительно максимум 7 из пар оснований в области дцРНК являются неканоническими парами оснований, и наиболее предпочтительно максимум 6 из пар оснований в области дцРНК являются неканоническими парами оснований. Предпочтительно в вышеуказанных вариантах осуществления неканонические пары оснований включают по меньшей мере одну пару оснований G:U, более предпочтительно все неканонические пары оснований являются парами оснований G:U. Предпочтительно признаки вышеуказанных вариантов осуществления относятся ко всем из 20 последовательных пар оснований, присутствующих в молекуле РНК согласно изобретению.
В варианте осуществления область дцРНК молекулы РНК изобретения включает 21 последовательную пару оснований, где по меньшей мере одна пара оснований из 21 последовательной пары оснований является неканонической парой оснований. В варианте осуществления область дцРНК включает 21 последовательную пару оснований, где по меньшей мере 2 пары оснований из 21 последовательной пары оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 21 последовательную пару оснований, где по меньшей мере 3 пары оснований из 21 последовательной пары оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 21 последовательную пару оснований, где по меньшей мере 4 пары оснований из 21 последовательной пары оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 21 последовательную пару оснований, где по меньшей мере 5 пар оснований из 21 последовательной пары оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 21 последовательную пару оснований, где по меньшей мере 6 пар оснований из 21 последовательной пары оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 21 последовательную пару оснований, где по меньшей мере 7 пар оснований из 21 последовательной пары оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 21 последовательную пару оснований, где по меньшей мере 8 пар оснований из 21 последовательной пары оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 21 последовательную пару оснований, где по меньшей мере 9 пар оснований из 21 последовательной пары оснований являются неканоническими парами оснований. В каждом из этих вариантов осуществления предпочтительно, что максимум 10 из 21 последовательной пары оснований в области дцРНК являются неканоническими парами оснований, более предпочтительно максимум 9 из пар оснований в области дцРНК являются неканоническими парами оснований, еще более предпочтительно максимум 8 из пар оснований в области дцРНК являются неканоническими парами оснований, даже еще более предпочтительно максимум 7 из пар оснований в области дцРНК являются неканоническими парами оснований, и наиболее предпочтительно максимум 6 из пар оснований в области дцРНК являются неканоническими парами оснований. Предпочтительно в вышеуказанных вариантах осуществления неканонические пары оснований включают по меньшей мере одну пару оснований G:U, более предпочтительно все неканонические пары оснований являются парами оснований G:U. Предпочтительно признаки вышеуказанных вариантов осуществления относятся ко всем из 21 последовательной пары оснований, присутствующих в молекуле РНК согласно изобретению.
В варианте осуществления область дцРНК молекулы РНК согласно изобретению включает 22 последовательных пары оснований, где по меньшей мере одна пара оснований из 22 последовательных пар оснований является неканонической парой оснований. В варианте осуществления область дцРНК включает 22 последовательных пары оснований, где по меньшей мере 2 пары оснований из 22 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 22 последовательных пары оснований, где по меньшей мере 3 пары оснований из 22 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 22 последовательных пары оснований, где по меньшей мере 4 пары оснований из 22 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 22 последовательных пары оснований, где по меньшей мере 5 пар оснований из 22 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 22 последовательных пары оснований, где по меньшей мере 6 пар оснований из 22 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 22 последовательных пары оснований, где по меньшей мере 7 пар оснований из 22 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 22 последовательных пары оснований, где по меньшей мере 8 пар оснований из 22 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 22 последовательных пары оснований, где по меньшей мере 9 пар оснований из 22 последовательных пар оснований являются неканоническими парами оснований. В каждом из этих вариантов осуществления предпочтительно, что максимум 10 из 22 последовательных пар оснований в области дцРНК являются неканоническими парами оснований, более предпочтительно максимум 9 из пар оснований в области дцРНК являются неканоническими парами оснований, еще более предпочтительно максимум 8 из пар оснований в области дцРНК являются неканоническими парами оснований, даже еще более предпочтительно максимум 7 из пар оснований в области дцРНК являются неканоническими парами оснований, и наиболее предпочтительно максимум 6 из пар оснований в области дцРНК являются неканоническими парами оснований. Предпочтительно в вышеуказанных вариантах осуществления неканонические пары оснований включают по меньшей мере одну пару оснований G:U, более предпочтительно все неканонические пары оснований являются парами оснований G:U. Предпочтительно признаки вышеуказанных вариантов осуществления относятся ко всем из 22 последовательных пар оснований, присутствующих в молекуле РНК согласно изобретению.
В варианте осуществления область дцРНК молекулы РНК согласно изобретению включает 23 последовательных пары оснований, где по меньшей мере одна пара оснований из 23 последовательных пар оснований является неканонической парой оснований. В варианте осуществления область дцРНК включает 23 последовательных пары оснований, где по меньшей мере 2 пары оснований из 23 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 23 последовательных пары оснований, где по меньшей мере 3 пары оснований из 23 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 23 последовательных пары оснований, где по меньшей мере 4 пары оснований из 23 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 23 последовательных пары оснований, где по меньшей мере 5 пар оснований из 23 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 23 последовательных пары оснований, где по меньшей мере 6 пар оснований из 23 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 23 последовательных пары оснований, где по меньшей мере 7 пар оснований из 23 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 23 последовательных пары оснований, где по меньшей мере 8 пар оснований из 23 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 23 последовательных пары оснований, где по меньшей мере 9 пар оснований из 23 последовательных пар оснований являются неканоническими парами оснований. В каждом из этих вариантов осуществления предпочтительно, что максимум 10 из 23 последовательных пар оснований в области дцРНК являются неканоническими парами оснований, более предпочтительно максимум 9 из пар оснований в области дцРНК являются неканоническими парами оснований, еще более предпочтительно максимум 8 из пар оснований в области дцРНК являются неканоническими парами оснований, даже еще более предпочтительно максимум 7 из пар оснований в области дцРНК являются неканоническими парами оснований, и наиболее предпочтительно максимум 6 из пар оснований в области дцРНК являются неканоническими парами оснований. Предпочтительно в вышеуказанных вариантах осуществления неканонические пары оснований включают по меньшей мере одну пару оснований G:U, более предпочтительно все неканонические пары оснований являются парами оснований G:U. Предпочтительно признаки вышеуказанных вариантов осуществления относятся ко всем из 23 последовательных пар оснований, присутствующих в молекуле РНК согласно изобретению.
В варианте осуществления область дцРНК молекулы РНК согласно изобретению включает 24 последовательных пары оснований, где по меньшей мере одна пара оснований из 24 последовательных пар оснований является неканонической парой оснований. В варианте осуществления область дцРНК включает 24 последовательных пары оснований, где по меньшей мере 2 пары оснований из 24 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 24 последовательных пары оснований, где по меньшей мере 3 пары оснований из 24 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 24 последовательных пары оснований, где по меньшей мере 4 пары оснований из 24 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 24 последовательных пары оснований, где по меньшей мере 5 пар оснований из 24 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 24 последовательных пары оснований, где по меньшей мере 6 пар оснований из 24 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 24 последовательных пары оснований, где по меньшей мере 7 пар оснований из 24 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 24 последовательных пары оснований, где по меньшей мере 8 пар оснований из 24 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 24 последовательных пары оснований, где по меньшей мере 9 пар оснований из 24 последовательных пар оснований являются неканоническими парами оснований. В каждом из этих вариантов осуществления предпочтительно, что максимум 10 из 24 последовательных пар оснований в области дцРНК являются неканоническими парами оснований, более предпочтительно максимум 9 из пар оснований в области дцРНК являются неканоническими парами оснований, еще более предпочтительно максимум 8 из пар оснований в области дцРНК являются неканоническими парами оснований, даже еще более предпочтительно максимум 7 из пар оснований в области дцРНК являются неканоническими парами оснований, и наиболее предпочтительно максимум 6 из пар оснований в области дцРНК являются неканоническими парами оснований. Предпочтительно в вышеуказанных вариантах осуществления неканонические пары оснований включают по меньшей мере одну пару оснований G:U, более предпочтительно все неканонические пары оснований являются парами оснований G:U. Предпочтительно признаки вышеуказанных вариантов осуществления относятся ко всем из 24 последовательных пар оснований, присутствующих в молекуле РНК согласно изобретению.
В следующих вариантах осуществления полноразмерная область дцРНК (т.е. полная область дцРНК) молекулы РНК согласно изобретению рассматривается как контекст для признака, если присутствует только одна (непрерывная) область дцРНК, или для каждой из областей дцРНК молекулы РНК, если в молекуле РНК присутствуют две или более областей дцРНК. В варианте осуществления область дцРНК не включает 20 последовательных канонических пар оснований, т.е. каждая субобласть из 20 последовательных пар оснований включает по меньшей мере одну неканоническую пару оснований, предпочтительно по меньшей мере одну пару оснований G:U. В варианте осуществления область дцРНК не включает 19 последовательных канонических пар оснований. В варианте осуществления область дцРНК не включает 18 последовательных канонических пар оснований. В варианте осуществления область дцРНК не включает 17 последовательных канонических пар оснований. В варианте осуществления область дцРНК не включает 16 последовательных канонических пар оснований. В варианте осуществления область дцРНК не включает 15 последовательных канонических пар оснований. В варианте осуществления область дцРНК не включает 14 последовательных канонических пар оснований. В варианте осуществления область дцРНК не включает 13 последовательных канонических пар оснований. В варианте осуществления область дцРНК не включает 12 последовательных канонических пар оснований. В варианте осуществления область дцРНК не включает 11 последовательных канонических пар оснований. В варианте осуществления область дцРНК не включает 10 последовательных канонических пар оснований. В варианте осуществления область дцРНК не включает 9 последовательных канонических пар оснований. В варианте осуществления область дцРНК не включает 8 последовательных канонических пар оснований. В варианте осуществления область дцРНК не включает 7 последовательных канонических пар оснований. В вышеуказанных вариантах осуществления предпочтительно, что наиболее протяженная непрерывная субобласть канонических спаренных оснований в области дцРНК молекулы РНК или каждой области дцРНК в молекуле РНК содержит 5, 6 или 7 последовательных канонических пар оснований, т.е. в направлении более коротких указанных длин. Каждый из признаков вышеуказанных вариантов осуществления предпочтительно объединен в молекуле РНК со следующими признаками. В варианте осуществления область дцРНК включает от 10 до 19 или 20 последовательных пар оснований. В предпочтительном варианте осуществления область дцРНК включает от 12 до 19 или 20 последовательных пар оснований. В варианте осуществления область дцРНК включает от 14 до 19 или 20 последовательных пар оснований. В этих вариантах осуществления область дцРНК включает 15 последовательных пар оснований. В варианте осуществления область дцРНК включает 16, 17, 18 или 19 последовательных пар оснований. В варианте осуществления область дцРНК включает 20 последовательных пар оснований. Предпочтительно в вышеуказанных вариантах осуществления последовательные пары оснований включают по меньшей мере одну неканоническую пару оснований, включающую по меньшей мере одну пару оснований G:U, более предпочтительно все неканонические пары оснований в области последовательных пар оснований являются парами оснований G:U.
В варианте осуществления область дцРНК включает субобласть из 4 канонических пар оснований между неканоническими парами оснований, т.е. по меньшей мере одна, предпочтительно одна или две (не больше 2) неканонические пары оснований примыкают к каждому концу 4 канонических пар оснований. В варианте осуществления область дцРНК включает 2 субобласти, по 4 канонических пары оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает 3 субобласти, по 4 канонических пары оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает 4 или 5 субобластей, по 4 канонических пары оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает 6 или 7 субобластей, по 4 канонических пары оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает 8-10 субобластей, по 4 канонических пары оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает 11-15 субобластей, по 4 канонических пары оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает от 2 до 50 субобластей, по 4 канонических пары оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает от 2 до 40 субобластей, по 4 канонических пары оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает от 2 до 30 субобластей, по 4 канонических пары оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает от 2 до 20 субобластей, по 4 канонических пары оснований каждая, между неканоническими парами оснований. Предпочтительно в вышеуказанных вариантах осуществления неканонические пары оснований включают по меньшей мере одну пару оснований G:U, более предпочтительно все неканонические пары оснований, примыкающие к последовательным каноническим парам оснований в субобластях, являются парами оснований G:U. В вариациях вышеуказанных вариантов осуществления одна или обе фланкирующих неканонических пар оснований заменены неспаренным рибонуклеотидом в смысловой последовательности, антисмысловой последовательности или в обеих последовательностях, в некоторых или всех субобластях. Должно быть очевидным, что в вышеуказанных вариантах осуществления максимальное количество субобластей определяется длиной области дцРНК в молекуле РНК.
В варианте осуществления область дцРНК включает субобласть из 5 канонических пар оснований между неканоническими парами оснований. В варианте осуществления область дцРНК включает 2 субобласти, по 5 канонических пар оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает 3 субобласти, по 5 канонических пар оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает 4 или 5 субобластей, по 5 канонических пар оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает 6 или 7 субобластей, по 5 канонических пар оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает 8-10 субобластей, по 5 канонических пар оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает 11-15 субобластей, по 5 канонических пар оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает от 2 до 50 субобластей, по 5 канонических пар оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает от 2 до 50 субобластей, по 5 канонических пар оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает от 2 до 30 субобластей, по 5 канонических пар оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает от 2 до 20 субобластей, по 5 канонических пар оснований каждая, между неканоническими парами оснований. Предпочтительно в вышеуказанных вариантах осуществления неканонические пары оснований включают по меньшей мере одну пару оснований G:U, более предпочтительно все неканонические пары оснований, примыкающие к смежным каноническим парам оснований в субобластях, являются парами оснований G:U. В вариациях вышеуказанных вариантов осуществления одна или обе фланкирующих неканонических пары оснований заменены неспаренным рибонуклеотидом в смысловой последовательности, антисмысловой последовательности или в обеих последовательностях, в некоторых или всех субобластях. Должно быть очевидным, что в вышеуказанных вариантах осуществления максимальное количество субобластей определяется длиной области дцРНК в молекуле РНК.
В варианте осуществления область дцРНК включает субобласть из 6 канонических пар оснований между неканоническими парами оснований. В варианте осуществления область дцРНК включает 2 субобласти, по 6 канонических пар оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает 3 субобласти, по 6 канонических пар оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает 4 или 5 субобластей, по 6 канонических пар оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает 6 или 7 субобластей, по 6 канонических пар оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает 8-10 субобластей, по 6 канонических пар оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает 11-16 субобластей, по 6 канонических пар оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает от 2 до 60 субобластей, по 6 канонических пар оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает от 2 до 60 субобластей, по 6 канонических пар оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает от 2 до 30 субобластей, по 6 канонических пар оснований каждая, между неканоническими парами оснований. В варианте осуществления область дцРНК включает от 2 до 20 субобластей, по 6 канонических пар оснований каждая, между неканоническими парами оснований. Предпочтительно в вышеуказанных вариантах осуществления неканонические пары оснований включают по меньшей мере одну пару оснований G:U, более предпочтительно все неканонические пары оснований, примыкающие к смежным каноническим парам оснований в субобластях, являются парами оснований G:U. В вариациях вышеуказанных вариантов осуществления одна или обе фланкирующих неканонических пары оснований заменены неспаренным рибонуклеотидом в смысловой последовательности, антисмысловой последовательности или в обеих последовательностях, в некоторых или всех субобластях. Должно быть очевидным, что в вышеуказанных вариантах осуществления максимальное количество субобластей определяется длиной области дцРНК в молекуле РНК.
В варианте осуществления область дцРНК включает субобласть из 10 последовательных пар оснований, где 2-4 из пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 2 субобласти, по 10 последовательных пар оснований каждая, где 2-4 из 10 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 3 субобласти, по 10 последовательных пар оснований каждая, где 2-4 из 10 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 4 субобласти, по 10 последовательных пар оснований каждая, где 2-4 из 10 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 5 субобластей, по 10 последовательных пар оснований каждая, где 2-4 из 10 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 10 субобластей, по 10 последовательных пар оснований каждая, где 2-4 из 10 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает 4 субобласти, по 15 последовательных пар оснований каждая, где 2-6 из 15 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает от 2 до 50 субобластей, по 10 последовательных пар оснований каждая, где 2-4 из 10 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает от 2 до 40 субобластей, по 10 последовательных пар оснований каждая, где 2-4 из 10 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает от 2 до 30 субобластей, по 10 последовательных пар оснований каждая, где 2-4 из 10 последовательных пар оснований являются неканоническими парами оснований. В варианте осуществления область дцРНК включает от 2 до 20 субобластей, по 10 последовательных пар оснований каждая, где 2-4 из 10 последовательных пар оснований являются неканоническими парами оснований. Предпочтительно в вышеуказанных вариантах осуществления неканонические пары оснований включают по меньшей мере одну пару оснований G:U, более предпочтительно все неканонические пары оснований в субобластях являются парами оснований G:U. В вариациях вышеуказанных вариантов осуществления одна или более из 2-4 или 2-6 неканонических пар оснований заменены неспаренным рибонуклеотидом в смысловой последовательности, антисмысловой последовательности или в обеих последовательностях, в некоторых или всех субобластях. Должно быть очевидным, что в вышеуказанных вариантах осуществления максимальное количество субобластей определяется длиной области дцРНК в молекуле РНК.
В варианте осуществления отношение канонических к неканоническим парам оснований в области дцРНК составляет от 2,5:1 до 3,5:1, например, приблизительно 3:1. В варианте осуществления отношение канонических к неканоническим парам оснований в области дцРНК составляет от 3,5:1 до 4.5:1, например, приблизительно 4:1. В варианте осуществления отношение канонических к неканоническим парам оснований в области дцРНК составляет от 4,5:1 до 5,5:1, например, приблизительно 5:1. В варианте осуществления отношение канонических к неканоническим парам оснований в области дцРНК составляет от 5,5:1 до 6,5:1, например, приблизительно 6:1. Различные области дцРНК в молекуле РНК могут иметь разные соотношения.
В вышеуказанных вариантах осуществления неканонические пары оснований в области(ях) дцРНК молекулы РНК предпочтительно все являются парами оснований G:U. В варианте осуществления по меньшей мере 99% неканонических пар оснований являются парами оснований G:U. В варианте осуществления по меньшей мере 98% неканонических пар оснований являются парами оснований G:U. В варианте осуществления по меньшей мере 97% неканонических пар оснований являются парами оснований G:U. В варианте осуществления по меньшей мере 95% неканонических пар оснований являются парами оснований G:U. В варианте осуществления по меньшей мере 90% неканонических пар оснований являются парами оснований G:U. В варианте осуществления 90-95% неканонических пар оснований являются парами оснований G:U. Например, в случае присутствия 10 неканонических пар оснований, по меньшей мере 9 (90%) являются парами оснований G:U.
Область дцРНК, включающая неканоническое спаренное основание(я), включает антисмысловую последовательность из 20 последовательных нуклеотидов, которая действует как антисмысловой регуляторный элемент. В варианте осуществления антисмысловой регуляторный элемент по меньшей мере на 80%, предпочтительно по меньшей мере на 90%, более предпочтительно по меньшей мере на 95% или наиболее предпочтительно на 100% комплементарен молекуле РНК-мишени в эукариотической клетке. В варианте осуществления область дцРНК включает 2, 3, 4 или 5 антисмысловых регуляторных элементов, которые либо комплементарны одной и той же молекуле РНК-мишени (т.е. различным областям одной молекулы РНК-мишени), либо комплементарны разным молекулам РНК-мишеней.
В варианте осуществления один или более рибонуклеотидов смысловой рибонуклеотидной последовательности и/или один или более рибонуклеотидов антисмысловой рибонуклеотидной последовательности не спарены в области дцРНК, когда смысловая и антисмысловая последовательности гибридизуются. В этом варианте осуществления область дцРНК не включает последовательность петли, которая ковалентно соединяет смысловую и антисмысловую последовательности. Один или более рибонуклеотидов области дцРНК или субобласти могут быть не спарены. Таким образом, в этом варианте осуществления смысловая цепь области дцРНК не спаривается полностью со своей соответствующей антисмысловой цепью.
Кроме того, в варианте осуществления и, необязательно, в комбинации с любым из признаков вышеуказанных вариантов осуществления общее количество рибонуклеотидов в смысловой последовательности(ях) и общее количество рибонуклеотидов в антисмысловой последовательности(ях) могут не быть идентичными, несмотря на то, что предпочтительно они идентичны. В варианте осуществления общее количество рибонуклеотидов в смысловой рибонуклеотидной последовательности(ях) области дцРНК составляет от 90% до 110% от общего количества рибонуклеотидов в антисмысловой рибонуклеотидной последовательности(ях). В варианте осуществления общее количество рибонуклеотидов в смысловой рибонуклеотидной последовательности(ях) составляет от 95% до 105% от общего количества рибонуклеотидов в антисмысловой рибонуклеотидной последовательности(ях). В варианте осуществления химерные молекулы РНК согласно настоящему изобретению могут включать один или более структурных элементов, таких как внутренние или концевые выпетливания или петли. Различные варианты осуществления выпетливаний и петель обсуждаются выше. В варианте осуществления области дцРНК отделены структурным элементом, таким как выпетливание или петля. В варианте осуществления, области дцРНК отделены промежуточной (спейсерной) последовательностью. Некоторые рибонуклеотиды спейсерной последовательности могут быть спарены с другими рибонуклеотидами в молекуле РНК, например, с другими рибонуклеотидами в спейсерной последовательности, или они могут быть не спарены в молекуле РНК или в некоторой части каждого. В варианте осуществления области дцРНК соединены с концевой петлей. В варианте осуществления области дцРНК фланкированы концевыми петлями.
В варианте осуществления, где область дцРНК молекулы РНК согласно изобретению содержит по меньшей мере 3 неканонических пары оснований в любой субобласти из 5 последовательных пар оснований, неканонические пары оснований не являются смежными, но отделены одной или более каноническими парами оснований, т.е. область дцРНК не содержит 3 или более последовательных неканонических пар оснований. В варианте осуществления область дцРНК не содержит 4 или более последовательных неканонических пар оснований. Например, в варианте осуществления область дцРНК включает по меньшей мере 3 неканонических пары оснований в субобласти из 10 пар оснований, где каждая неканоническая пара оснований отделена 4 каноническими парами оснований.
В варианте осуществления молекула РНК согласно изобретению включает больше одной области дцРНК. Например, молекула РНК включает 2, 3, 4, 5, 6, 7, 8, 9, 10 или больше областей дцРНК. В этом примере одна или больше областей, или все области дцРНК могут включать представленные выше свойства, такие как неканоническое спаривание оснований и/или количество антисмысловых регуляторных элементов.
Сайленсирующая активность
Молекулы РНК согласно настоящему изобретению обладают антисмысловой активностью, так как они содержат смысловую рибонуклеотидную последовательность, которая по существу комплементарна области молекулы РНК-мишени. Например, рибонуклеотидная последовательность по существу комплементарна области молекулы РНК-мишени в эукариотической клетке. В одном примере молекула РНК-мишени может находиться в клетке бактерии, клетке гриба, клетке растения, клетке насекомого или клетке животного. Такие компоненты молекул РНК, определенные в настоящем документе, могут именоваться "антисмысловым регуляторным элементом". "По существу комплементарный" означает, что смысловая рибонуклеотидная последовательность может иметь вставки, делеции и отдельные точечные мутации по сравнению с комплементом молекулы РНК-мишени в эукариотической клетке. Предпочтительно гомология между смысловой рибонуклеотидной последовательностью с антисмысловой активностью и молекулой РНК-мишени составляет по меньшей мере 80%, предпочтительно по меньшей мере 90%, предпочтительно по меньшей мере 95%, наиболее предпочтительно 100%. Например, смысловая рибонуклеотидная последовательность может включать приблизительно 15, приблизительно 16, приблизительно 17, приблизительно 18, приблизительно 19 или больше последовательных нуклеотидов, которые идентичны по последовательности первой области молекулы РНК-мишени в эукариотической клетке. В другом примере смысловая рибонуклеотидная последовательность может включать приблизительно 20 последовательных нуклеотидов, которые идентичны по последовательности первой области молекулы РНК-мишени в эукариотической клетке.
"Антисмысловая активность" используется в рамках настоящего описания для обозначения антисмыслового регуляторного элемента из молекулы РНК, определенной в настоящем документе, который модулирует (увеличивает или уменьшает) экспрессию молекулы РНК-мишени.
В различных примерах антисмысловые регуляторные элементы согласно настоящему изобретению могут включать множество мономерных субъединиц, соединенных вместе соединительными группами. Примеры включают праймеры, зонды, антисмысловые соединения, антисмысловые олигонуклеотиды, олигонуклеотиды с внешней направляющей последовательностью (EGS), альтернативные сплайсеры, гапмеры, миРНК и микроРНК. В таком качестве молекулы РНК согласно настоящему раскрытию могут содержать антисмысловые регуляторные элементы с одноцепочечными, двухцепочечными, кольцевыми, разветвленными или шпилечными структурами. В одном примере антисмысловая последовательность может содержать структурные элементы, такие как внутренние или концевые выпетливания или петли.
В примере молекулы РНК согласно настоящему изобретению включают химерные олигомерные компоненты, такие как химерные олигонуклеотиды. Например, молекула РНК может включать различные модифицированные нуклеотиды, антисмысловые олигонуклеотиды со смешанным скелетом или их комбинацию. В примере химерные олигомерные соединения могут включать по меньшей мере одну область, модифицированную с обеспечением повышенной устойчивости к расщеплению нуклеазами, повышенного клеточного захвата и/или повышенной аффинности связывания с молекулой РНК-мишени.
Антисмысловые регуляторные элементы могут иметь различную длину. В различных примерах настоящего изобретения предложены антисмысловые регуляторные элементы, состоящие из X-Y связанных оснований, где каждый X и Y независимо выбран из 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49 и 50 (при условии, что X<Y). Например, в некоторых вариантах осуществления настоящего изобретения предложены антисмысловые регуляторные элементы, включающие: 8-9, 8-10, 8-11, 8-12, 8-13, 8-14, 8-15, 8-16, 8-17, 8-18, 8-19, 8-20, 8-21, 8-22, 8-23, 8-24, 8-25, 8-26, 8-27, 8-28, 8-29, 8-30, 9-10, 9-11, 9-12, 9-13, 9-14, 9-15, 9-16, 9-17, 9-18, 9-19, 9-20, 9-21, 9-22, 9-23, 9-24, 9-25, 9-26, 9-27, 9-28, 9-29, 9-30, 10-11, 10-12, 10-13, 10-14, 10-15, 10-16, 10-17, 10-18, 10-19, 10-20, 10-21, 10-22, 10-23, 10-24, 10-25, 10-26, 10-27, 10-28, 10-29, 10-30, 11-12, 11-13, 11-14, 11-15, 11-16, 11-17, 11-18, 11-19, 11-20, 11-21, 11-22, 11-23, 11-24, 11-25, 11-26, 11-27, 11-28, 11-29, 11-30, 12-13, 12-14, 12-15, 12-16, 12-17, 12-18, 12-19, 12-20, 12-21, 12-22, 12-23, 12-24, 12-25, 12-26, 12-27, 12-28, 12-29, 12-30, 13-14, 13-15, 13-16, 13-17, 13-18, 13-19, 13-20, 13-21, 13-22, 13-23, 13-24, 13-25, 13-26, 13-27, 13-28, 13-29, 13-30, 14-15, 14-16, 14-17, 14-18, 14-19, 14-20, 14-21, 14-22, 14-23, 14-24, 14-25, 14-26, 14-27, 14-28, 14-29, 14-30, 15-16, 15-17, 15-18, 15-19, 15-20, 15-21, 15-22, 15-23, 15-24, 15-25, 15-26, 15-27, 15-28, 15-29, 15-30, 16-17, 16-18, 16-19, 16-20, 16-21, 16-22, 16-23, 16-24, 16-25, 16-26, 16-27, 16-28, 16-29, 16-30, 17-18, 17-19, 17-20, 17-21, 17-22, 17-23, 17-24, 17-25, 17-26, 17-27, 17-28, 17-29, 17-30, 18-19, 18-20, 18-21, 18-22, 18-23, 18-24, 18-25, 18-26, 18-27, 18-28, 18-29, 18-30, 19-20, 19-21, 19-22, 19-23, 19-24, 19-25, 19-26, 19-29, 19-28, 19-29, 19-30, 20-21, 20-22, 20-23, 20-24, 20-25, 20-26, 20-27, 20-28, 20-29, 20-30, 21-22, 21-23, 21-24, 21-25, 21-26, 21-27, 21-28, 21-29, 21-30, 22-23, 22-24, 22-25, 22-26, 22-27, 22-28, 22-29, 22-30, 23-24, 23-25, 23-26, 23-27, 23-28, 23-29, 23-30, 24-25, 24-26, 24-27, 24-28, 24-29, 24-30, 25-26, 25-27, 25-28, 25-29, 25-30, 26-27, 26-28, 26-29, 26-30, 27-28, 27-29, 27-30, 28-29, 28-30 или 29-30 связанных оснований.
Молекулы РНК согласно настоящему изобретению могут включать множество антисмысловых регуляторных элементов. Например, молекулы РНК могут включать по меньшей мере 2, по меньшей мере 3, по меньшей мере 4, по меньшей мере 5, по меньшей мере 6, по меньшей мере 7, по меньшей мере 8, по меньшей мере 9, по меньшей мере 10 антисмысловых регуляторных элементов. В примере антисмысловые регуляторные элементы являются одинаковыми. В этом примере молекула РНК может включать по меньшей мере 2, по меньшей мере 3, по меньшей мере 4, по меньшей мере 5, по меньшей мере 6, по меньшей мере 7, по меньшей мере 8, по меньшей мере 9, по меньшей мере 10 копий антисмыслового регулирующего элемента. В другом примере молекулы РНК согласно настоящему изобретению могут включать разные антисмысловые регуляторные элементы. Например, антисмысловые регуляторные элементы могут быть предоставлены для направленного воздействия на множество генов в метаболическом пути, таком как биосинтез липидов. В этом примере молекула РНК может включать по меньшей мере 2, по меньшей мере 3, по меньшей мере 4, по меньшей мере 5, по меньшей мере 6, по меньшей мере 7, по меньшей мере 8, по меньшей мере 9, по меньшей мере 10 разных антисмысловых регуляторных элементов.
Антисмысловые регуляторные элементы согласно настоящему изобретению могут модулировать (увеличивать или уменьшать) экспрессию или количество различных молекул РНК-мишеней. В примере молекула РНК-мишени является геном биосинтеза жирной кислоты. Примеры таких генов включают ген, кодирующий ацетилтрансацилазы, ацилпереносящие белки ("белок-переносчик ацила"), десатуразы, такие как стеарилдесатуразы или микросомальные D12-десатуразы, в частности гены Fad2-1, малонилтрансацилазу, -кетоацил-ACP-синтетазы, 3- кето-ACP-редуктазы, еноил-ACP-гидразы, тиоэстеразы, такие как ацил-ACP-тиоэстеразы, еноил-ACP-редуктазы. В одном примере молекулой РНК-мишени является ген FAD2 (например, который описан в Genbank под рег. номерами AF124360 (Brassica carinata), AF042841 (Brassica rapa), L26296 (Arabidopsis thaliana), A65102 (Corylus avellana)). Например, молекулой РНК-мишени может быть ген FAD2.1. В другом примере молекулой РНК-мишени может быть ген FAD2.2. В другом примере молекулой РНК-мишени могут быть гены FAD2.1 и FAD2.2. Примеры других генов, участвующих в модификации липидного состава, которые могут быть молекулой РНК-мишени, известны в уровне техники (Shure M et al. (1983) Cell 35:225-233; Preiss et al. (1987) Tailoring Genes for Crop Improvement (Bruening et al., eds.), Plenum Press, S.133-152; Gupta et al. (1988) Plant Mol. Biol. 10:215-224; Olive et al. (1989) Plant Mol Biol 12:525-538; Bhattacharyya et al. (1990) Cell 60:155-122; Dunwell J M (2000) J Exp Botany 51Spec No:487-96; Brar D S et al. (1996) Biotech Genet. Eng Rev 13:167-79; Kishore G M and Somerville C R (1993) Curr Opin Biotech 4(2):152-8; US 5,530,192 и WO 94/18337).
В другом примере молекулой РНК-мишени является ген членистоногого, такой как транскрипт гена насекомого. Примеры таких генов включают гены хитинсинтазы, такие как CHS1 и/или CHS2, или другие гены, регулирующие активность, поведение, воспроизводство, рост и/или развитие насекомого. Различные важные гены множества патогенов известны специалистам в данной области (например, гены устойчивости к нематодам приведены в WO 93/10251, WO 94/17194).
В другом примере молекула РНК-мишени ассоциирована с заболеванием. Например, молекула РНК-мишени может быть транскриптом онкогена или гена-супрессора опухоли. Примеры онкогенов включают ABL1, BCL1, BCL2, BCL6, CBFA2, CBL, CSF1R, ERBA, ERBB, EBRB2, FGR, FOS, FYN, HRAS, JUN, LCK, LYN, MYB, MYC, NRAS, RET или SRC. Примеры генов-супрессоров опухоли включают BRCA1 или BRCA2; молекулы адгезии; циклинзависимые киназы и их ингибиторы.
В другом примере молекула РНК-мишени связана с задержкой созревания плода растения. Задержка созревания плода растения может быть достигнута, например, путем снижения экспрессии генов, выбранных из группы, состоящей из полигалактуроназ, пектинэстераз, β-(1-4)глюканаз (целлюлаз), β-галактаназ (β-галактозидаз) или генов биосинтеза этилена, таких как 1-аминоциклопропан-1-карбоксилатсинтаза, гены биосинтеза каротиноидов, такие как, например, гены биосинтеза префитоена или фитоена, например фитоендесатуразы.
В другом примере молекула РНК-мишени связана с задержкой симптомов старения. Подходящие молекулы РНК-мишеней включают циннамоил-КоА:НАДФН-редуктазы или циннамоил-алкогольдегидрогеназу. Другие молекулы РНК-мишеней описаны (в WO 1995/07993).
В другом примере молекула РНК-мишени связана с модификацией содержания волокна в продуктах питания, предпочтительно в семенах. Например, молекула РНК может снижать экспрессию O-метилтрансферазы кофейной кислоты или циннамоил-алкогольдегидрогеназы.
Нуклеиновые кислоты, кодирующие молекулы РНК
Специалист в данной области поймет из представленного выше описания, что в настоящем изобретении также предложена выделенная нуклеиновая кислота, кодирующая молекулы РНК, раскрытые в настоящем документе, и их компоненты. Например, нуклеиновая кислота, включающая последовательность, представленную в любой одной или более из SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9. Нуклеиновая кислота может быть частично очищена после экспрессии в клетке-хозяине. Термин "частично очищенный" используется для обозначения молекулы РНК, которая обычно отделена от липидов, нуклеиновых кислот, других пептидов и других контаминирующих молекул, с которыми она ассоциирована в клетке-хозяине. Предпочтительно частично очищенный полинуклеотид очищен по меньшей мере на 60%, более предпочтительно по меньшей мере на 75% и более предпочтительно по меньшей мере на 90% от других компонентов, с которыми он ассоциирован.
В другом примере полинуклеотид согласно настоящему изобретению является гетерологичным полинуклеотидом. Термин "гетерологичный полинуклеотид" хорошо известен в уровне техники и относится к полинуклеотиду, который не является эндогенным по отношению к клетке или является нативным полинуклеотидом, в котором нативная последовательность была изменена, или нативным полипептидом, экспрессия которого изменена количественно в результате манипуляции с клеткой при помощи методов генной инженерии.
В другом примере полинуклеотид согласно настоящему изобретению является синтетическим полинуклеотидом. Например, полинуклеотид может быть получен с помощью методов, не требующих существования исходных последовательностей нуклеиновых кислот, таких как печать ДНК и синтез олигонуклеотидов. В другом примере полинуклеотид получен из ксенонуклеиновых кислот.
В примере полинуклеотид, раскрытый в настоящем документе, кодирует молекулу РНК-предшественника, включающую интрон по меньшей мере в одной последовательности петли, который может быть удален при транскрипции полинуклеотида в клетке-хозяине. В другом примере последовательность петли включает два, три, четыре, пять или больше интронов. В настоящем изобретении также предложена экспрессионная конструкцию, такая как конструкция ДНК, включающая выделенную нуклеиновую кислоту согласно изобретению, функционально связанную с промотором. В примере такие выделенные нуклеиновые кислоты и/или экспрессионные конструкции предоставлены в клетке или не относящемся к человеку организме. В примерах выделенные нуклеиновые кислоты стабильно интегрированы в геном клетки или не относящегося к человеку организма. Различные примеры подходящих экспрессионных конструкций, промоторов и клеток, включающих их, обсуждаются ниже.
Синтез молекул РНК согласно настоящему изобретению может быть выполнен с помощью различных методов, известных в уровне техники. В разделе Примеры представлен пример синтеза in vitro. В этом примере конструкции, включающие молекулы РНК, раскрытые в настоящем документе, расщепляют на 3'-конце, осаждают, очищают и подвергают количественному анализу. Синтез РНК может быть выполнен в бактериальной культуре после трансформации электро-компетентных клеток HT115 и индукции синтеза РНК с использованием T7, IPTG системы.
Рекомбинантные векторы
Один из вариантов осуществления настоящего изобретения включает рекомбинантный вектор, который содержит по меньшей мере одну молекулу РНК, определенную в настоящем документе, и способен доставлять молекулу РНК в клетку-хозяина. Рекомбинантные векторы включают векторы экспрессии. Рекомбинантные векторы содержат гетерологичные полинуклеотидные последовательности, то есть полинуклеотидные последовательности, которые не встречаются в природе в контакте с молекулой РНК, определенной в настоящем документе, которые предпочтительно происходят из разных видов. Вектор может представлять собой либо РНК, либо ДНК и, как правило, является вирусным вектором, полученным из вируса, или плазмидой.
Различные вирусные векторы могут использоваться для доставки и опосредования экспрессии молекулы РНК согласно настоящему изобретению. Выбор вирусного вектора, как правило, будет зависеть от различных параметров, таких как клетка или ткань, являющиеся мишенью для доставки, эффективность трансдукции вектора и патогенность. В одном примере вирусный вектор интегрируется в хозяйский клеточный хроматин (например, лентивирусы). В другом примере вирусный вектор сохраняется в ядре клетки преимущественно в виде внехромосомной эписомы (например, аденовирусы). Примеры этих типов вирусных векторов включают онкоретровирусы, лентивирусы, аденоассоциированные вирусы, аденовирусы, вирусы герпеса и ретровирусы.
Плазмидные векторы обычно включают дополнительные последовательности нуклеиновых кислот, которые обеспечивают легкий отбор, амплификацию и трансформацию кассеты экспрессии в прокариотических клетках, например, векторы на основе pUC, векторы на основе pGEM или бинарные векторы, содержащие одну или несколько областей Т-ДНК. Дополнительные последовательности нуклеиновых кислот включают точки начала репликации для обеспечения автономной репликации вектора, селективные маркерные гены, предпочтительно кодирующие устойчивость к антибиотикам или гербицидам, уникальные сайты множественного клонирования, обеспечивающие множественные сайты для вставки последовательностей нуклеиновых кислот или генов, кодируемых в конструкции нуклеиновой кислоты, и последовательности, которые усиливают трансформацию прокариотических и эукариотических (особенно растительных) клеток.
"Функционально связанный" при использовании в настоящем документе относится к функциональному отношению между двумя или более сегментами нуклеиновых кислот (например, ДНК). Как правило, это относится к функциональному отношению элемента регуляции транскрипции (промотора) к транскрибируемой последовательности. Например, промотор функционально связан с кодирующей последовательностью молекулы РНК, определенной в настоящем документе, если он стимулирует или модулирует транскрипцию кодирующей последовательности в подходящей клетке. Обычно промоторные элементы регуляции транскрипции, функционально связанные с транскрибируемой последовательностью, физически примыкают к транскрибируемой последовательности, т.е. они являются цис-действующими. Однако некоторые элементы регуляции транскрипции, такие как энхансеры, не должны физически примыкать или располагаться в непосредственной близости от кодирующих последовательностей, транскрипцию которых они усиливают.
Если присутствует множество промоторов, промоторы могут быть независимо одинаковыми или разными.
Для облегчения идентификации трансформантов, рекомбинантный вектор предпочтительно содержит селективный или скринируемый маркерный ген. Под "маркерным геном" подразумевается ген, который придает клеткам, экспрессирующим маркерный ген, определенный фенотип и, таким образом, позволяет отличать такие трансформированные клетки от клеток, которые не имеют маркера. Селективный маркерный ген придает признак, по которому можно "отбирать" на основе устойчивости к селективному агенту (например, гербициду, антибиотику). Скринируемый маркерный ген (или репортерный ген) придает признак, который можно идентифицировать с помощью визуального наблюдения или анализа, то есть путем "скрининга" (например, β-глюкуронидаза, люцифераза, GFP или другая ферментативная активность, отсутствующая в нетрансформированных клетках). Типичные селективные маркеры для отбора растительных трансформантов включают, без ограничения, ген hyg, который кодирует устойчивость к гигромицину B; ген неомицинфосфотрансферазы (nptII), придающий устойчивость к канамицину, паромомицину; ген глутатион-S-трансферазы из печени крысы, придающий устойчивость к гербицидам, полученным из глутатиона, как, например, описано в ЕР 256223; ген глутаминсинтетазы, придающий при оверэкспрессии устойчивость к ингибиторам глутаминсинтетазы, таким как фосфинотрицин, как, например, описано в WO 87/05327; ген ацетилтрансферазы из Streptomyces viridochromogenes, придающий устойчивость к селективному агенту фосфинотрицину, как, например, описано в ЕР 275957; ген, кодирующий 5-енолшикимат-3-фосфатсинтазу (EPSPS), придающий устойчивость к N-фосфонометилглицину, как, например, описано в публикации Hinchee et al. (1988); ген bar, придающий устойчивость к биалафосу, как, например, описано в WO91/02071; ген нитрилазы, такой как bxn из Klebsiella ozaenae, который придает устойчивость к бромоксинилу (Stalker et al., 1988); ген дигидрофолатредуктазы (DHFR), придающий устойчивость к метотрексату (Thillet et al., 1988); мутантный ген ацетолактатсинтазы (ALS), который придает устойчивость к имидазолинону, сульфонилмочевине или другим ингибирующим ALS химическим веществам (EP 154,204); мутантный ген антранилатсинтазы, который придает устойчивость к 5-метил-триптофану; или ген дегалогеназы, который придает устойчивость к гербициду далапону.
Предпочтительно рекомбинантный вектор стабильно включается в геном клетки, такой как клетка растения. Таким образом, рекомбинантный вектор может включать соответствующие элементы, которые позволяют вектору включаться в геном или в хромосому клетки.
Вектор экспрессии
При использовании в настоящем документе, "вектор экспрессии" является вектором ДНК, который способен трансформировать клетку-хозяина и вызывать экспрессию молекулы РНК, определенной в настоящем документе. Векторы экспрессии согласно настоящему изобретению содержат регуляторные последовательности, такие как последовательности контроля транскрипции, последовательности контроля трансляции, точки начала репликации и другие регуляторные последовательности, которые совместимы с клеткой-хозяином и которые регулируют экспрессию молекулы РНК согласно настоящему изобретению. В частности, векторы экспрессии согласно настоящему изобретению включают последовательности контроля транскрипции. Последовательности контроля транскрипции являются последовательностями, регулирующими инициацию, элонгацию и терминацию транскрипции. Особенно важными последовательностями контроля транскрипции являются последовательности, которые регулируют инициацию транскрипции, такие как последовательности промотора, энхансера, оператора и репрессора. Выбор используемых регуляторных последовательностей зависит от целевого организма, такого как растение, и/или целевого органа или представляющей интерес ткани. Такие регуляторные последовательности могут быть получены из любого эукариотического организма, такого как растения или вирусы растений, или могут быть химически синтезированы.
Примеры векторов, подходящие для стабильной трансфекции растительных клеток или для создания трансгенных растений, были описаны, например, в публикациях Pouwels et al., Cloning Vectors: A Laboratory Manual, 1985, supp. 1987, Weissbach and Weissbach, Methods for Plant Molecular Biology, Academic Press, 1989, и Gelvin et al., Plant Molecular Biology Manual, Kluwer Academic Publishers, 1990. Как правило, растительные векторы экспрессии включают, например, один или более клонированных генов растения под транскрипционным контролем 5'- и 3'-регуляторных последовательностей и доминантный селективный маркер. Такие векторы для экспрессии в растениях также могут содержать промоторную регуляторную область (например, регуляторную область, контролирующую индуцируемую или конститутивную, регулируемую условиями окружающей среды или регулируемую развитием, или клеточно- или тканеспецифическую экспрессию), сайт инициации транскрипции, сайт связывания рибосомы, сайт терминации транскрипции и/или сигнал полиаденилирования.
Векторы изобретения также могут применяться для получения молекул РНК, определенные в настоящем документе, в бесклеточной системе экспрессии, такие системы известны в уровне техники.
В примере полинуклеотид, кодирующий молекулу РНК согласно настоящему изобретению, функционально связан с промотором, способным к прямой экспрессии молекулы РНК в клетке-хозяине. В примере промотор функционирует in vitro. В примере промотор является промотором РНК-полимеразы. Например, промотор может быть промотором РНК-полимеразы III. В другом примере промотор может быть промотором РНК-полимеразы II. Однако выбор промотора может зависеть от целевого организма, такого как растение, насекомое, и/или целевого органа или представляющей интерес ткани. Примеры промоторов млекопитающих включают CMV, EF1a, SV40, PGK1, Ubc, человеческий бета-актин, CAG, TRE, UAS, CaMKIIa, CAL1, 10, TEF1, GDS, ADH1, CaMV35S, Ubi, H1 и U6. Примеры промоторов насекомых включают Ac5 и полиэдрин. Также был описан ряд конститутивных промоторов, которые активны в клетках растений. Подходящие промоторы для конститутивной экспрессии в растениях включают, без ограничения перечисленными, 35S промотор вирус мозаики цветной капусты (CaMV), 35S вируса мозаики норичника (FMV), фотоиндуцируемый промотор малой субъединицы (SSU) рибулозо-1,5-бисфосфаткарбоксилазы, промотор цитозольной триозофосфатизомеразы риса, промотор аденин- фосфорибозилтрансферазы Arabidopsis, промотор гена актина-1 риса, промоторы маннопинсинтазы и октопинсинтазы, промотор Adh, промотор сахарозосинтазы, промотор комплекса R-генов и промотор гена хлорофилл α/β-связывающего белка. Эти промоторы использовали для создания ДНК-векторов, экспрессируемых в растениях, см., например, WO 84/02913. Все эти промоторы использовали для создания различных типов экспрессируемых в растениях рекомбинантных ДНК-векторов.
В целях экспрессии в исходных тканях растения, таких как лист, семя, корень или стебель, предпочтительно, чтобы промоторы, применяемые в настоящем изобретении, имели относительно высокую экспрессию в этих определенных тканях. С этой целью можно сделать выбор из различных промоторов генов с тканевой или клеточной специфичностью или повышенной экспрессией. Примеры таких промоторов, описанные в литературе, включают, промотор глутамин-синтетазы GS2 хлоропластов из гороха, промотор фруктозо-1,6-бифосфатазы хлоропластов из пшеницы, ядерный фотосинтетический промотор ST-LS1 из картофеля, промотор серин/треонинкиназы и промотор глюкоамилазы (CHS) из Arabidopsis thaliana. Также описаны промоторы, активные в фотосинтетически активных тканях, такие как промотор рибулозо-1,5-бифосфаткарбоксилазы из лиственницы американской (Larix laricana), промотор гена Cab, Cab6, из сосны, промотор гена Cab-1 из пшеницы, промотор гена Cab-1 из шпината, промотор гена Cab 1R из риса, промотор пируватортофосфатдикиназы (PPDK) из Zea mays, промотор гена табака Lhcb1*2, промотор симпортера сахарозы-Н30 Suc2 Arabidopsis thaliana и промотор генов белков тилакоидной мембраны из шпината (PsaD, PsaF, PsaE, PC, FNR, AtpC, AtpD, Cab, RbcS). Другие промоторы хлорофилл α/β-связывающих белков также могут быть использоваться в настоящем изобретении, такие как промоторы гена LhcB и гена PsbP из горчицы белой (Sinapis alba).
Различные промоторы генов растений, регулируемые в ответ на сигналы внешней среды, гормональные, химические сигналы и/или сигналы развития, также могут использоваться для экспрессии генов РНК-связывающих белков в клетках растений, включая промоторы, регулируемые: (1) теплом, (2) светом (например, промотор RbcS-3А гороха, промотор RbcS кукурузы), (3) гормонами, такими как абсцизовая кислота, (4) повреждением (например, WunI) или (5) химическими веществами, такими как метилжасмонат, салициловая кислота, стероидные гормоны, спирт, антидоты гербицидов (WO 97/06269), или с преимуществами могут применяться (6) органоспецифические промоторы.
Используемый в настоящем документе термин "специфичный к запасающему органу растения промотор" относится к промотору, который предпочтительно, по сравнению с другими тканями растения, направляет транскрипцию гена в запасающем органе растения. Для экспрессии в запасающих тканях растения, таких как клубни картофеля, плоды томата или семена сои, канолы, хлопка, Zea mays, пшеницы, риса и ячменя предпочтительно, чтобы промоторы, применяемые в настоящем изобретении, обладали относительно высокой экспрессией в этих конкретных тканях. Может использоваться промотор β-конглицинина или другие семяспецифические промоторы, такие как промоторы напина, зеина, линина и фазеолина. Также могут использоваться корнеспецифические промоторы. Примером такого промотора является промотор гена кислой хитиназы. Экспрессия в корневой ткани также может осуществлятья при использовании корнеспецифических субдоменов 35S промотора CaMV, который был идентифицирован.
В особенно предпочтительном варианте осуществления промотор направляет экспрессию в тканях и органах, в которых происходит биосинтез липидов. Такие промоторы могут действовать при развитии семян в подходящее время для модификации липидного состава в семенах. Предпочтительные промоторы для семяспецифической экспрессии включают: 1) промоторы из генов, кодирующих ферменты, участвующие в биосинтезе и накоплении липидов в семенах, таких как десатуразы и элонгазы, 2) промоторы из генов, кодирующих запасные белки семян, и 3) промоторы из генов, кодирующих ферменты, участвующие в биосинтезе и накоплении углеводов в семенах. Подходящими семяспецифическими промоторами являются промотор гена напина рааса (US 5,608,152), промотор USP Vicia faba (Baumlein et al., 1991), промотор олеозина Arabidopsis (WO 98/45461), промотор фазеолина Phaseolus vulgaris (US 5,504,200), промотор Bce4 Brassica (WO 91/13980) или промотор легумина B4 (Baumlein et al., 1992), а также промоторы, которые приводят к семяспецифической экспрессии в однодольных растениях, таких как кукуруза, ячмень, пшеница, рожь, рис и т.п. Важными подходящими промоторами являются промотор гена lpt2 или lpt1 ячменя (WO 95/15389 и WO 95/23230) или промоторы, описанные в WO 99/16890 (промоторы из гена хордеина ячменя, гена глютелина риса, гена оризина риса, гена проламина риса, гена глиадина пшеницы, гена глютелина пшеницы, ген зеина кукурузы, гена глютелина овса, гена казирина сорго, гена секалина ржи). Другие промоторы включают промоторы, описанные в публикациях Broun et al. (1998), Potenza et al. (2004), US 20070192902 и US 20030159173. В одном варианте осуществления семяспецифический промотор предпочтительно экспрессируется в определенных частях семени, таких как семядоля(и) или эндосперм. Примеры семядоля-специфических промоторов включают, без ограничения перечисленными, промотор FP1 (Ellerstrom et al., 1996), промотор легумина гороха (Perrin et al., 2000) и промотор фитогемагглютинина фасоли (Perrin et al., 2000). Примеры эндосперм-специфических промоторов включают, без ограничения перечисленными, промотор зеина-1 кукурузы (Chikwamba et al., 2003), промотор глютелина-1 риса (Yang et al., 2003), промотор D-хордеина ячменя (Horvath et al., 2000) и промоторы глютелина HMW пшеницы (Alvarez et al., 2000). В другом варианте осуществления семяспецифический промотор не экспрессируется или экспрессируется лишь на низком уровне, в зародыше и/или после прорастания семени.
В другом варианте осуществления промотор, специфический для запасающего органа растения, является плодоспецифическим промотором. Примеры включают, без ограничения перечисленными, промоторы полигалактуроназы томата, Е8 и Pds, а также промотор оксидазы АСС яблок (см. обзор Potenza et al., 2004). В предпочтительном варианте осуществления промотор предпочтительно направляет экспрессию в съедобных частях плода, например, в мякоти плода, по сравнению с кожурой плода или с семенами внутри плода.
В одном варианте осуществления индуцируемый промотор представляет собой систему alc Aspergillus nidulans. Примеры индуцируемых систем экспрессии, которые могут использоваться вместо системы alc Aspergillus nidulans, описаны в обзорах Padidam (2003) и Corrado and Karali (2009). В другом варианте осуществления индуцируемый промотор является промотором, индуцируемым антидотом гербицида, таким как, например, промотор ln2-1 или ln2-2 кукурузы (Hershey and Stoner, 1991), промотор, индуцируемый антидотом гербицида, является промотором GST-27 кукурузы (Jepson et al., 1994) или промотором GH2/4 сои (Ulmasov et al., 1995).
В другом варианте осуществления индуцируемый промотор является индуцируемым старением промотором, таким как, например, индуцируемый старением промотор SAG (ассоциированный со старением ген) 12 и SAG 13 из Arabidopsis (Gan, 1995; Gan and Amasino, 1995) и LSC54 из Brassica napus (Buchanan-Wollaston, 1994). Такие промоторы демонстрируют повышенную экспрессию приблизительно при начале старения растительных тканей, в особенности листьев.
Для экспрессии в вегетационной ткани могут использоваться листоспецифические промоторы, такие как промоторы рибулозобифосфаткарбоксилазы (RBCS). Например, гены томата RBCS1, RBCS2 и RBCS3A экспрессируются в листьях и выращенной на свету рассаде (Meier et al., 1997). Могут использоваться промоторы рибулозобифосфаткарбоксилазы, экспрессируемой почти исключительно в мезофильных клетках пластин и влагалищ листьев в высоких концентрациях, описанные в публикации Matsuoka et al., (1994). Другим листоспецифическим промотором является промотор гена светособирающего хлорофилл a/b-связывающего белка (см. Shiina et al., 1997). Промотор myb-родственного гена Arabidopsis thaliana (Atmyb5), описанный в публикации Li et al., (1996), является листоспецифическим. Промотор Atmyb5 экспрессируется в развивающихся трихомах листьев, прилистниках и эпидермальных клетках и по краям молодых розеток и стеблевых листьев, а также в незрелых семенах. Также может использоваться промотор листьев, обнаруженный в кукурузе (Busk et al. (1997)).
В некоторых случаях, например, при рекомбинантной экспрессии LEC2 или BBM, может быть желательно, чтобы трансген не экспрессировался на высоких уровнях. Примером промотора, который может использоваться при таких обстоятельствах, является усеченный промотор напина A, который сохраняет профиль семяспецифической экспрессии, но со сниженным уровнем экспрессии (Tan et al., 2011).
5'-нетранслируемая лидерная последовательность может быть получена из промотора, выбранного для экспрессии последовательности гетерологичного гена молекулы РНК настоящего изобретения, или может быть гетерологичной в отношении кодирующей области продуцируемого фермента, и может быть специфически модифицирована при необходимости для повышения трансляции мРНК. С обзором по оптимизации экспрессии трансгенов можно ознакомиться в публикации Koziel et al. (1996). 5'-нетранслируемые области также могут быть получены из РНК вирусов растений (вируса табачной мозаики, вируса гравировки табака, вируса карликовой мозаики кукурузы, вируса мозаики люцерны и др.), из подходящих эукариотических генов, генов растений (лидерной последовательности из гена хлорофилл a/b-связывающего белка пшеницы и кукурузы) или из синтетических генных последовательностей. Настоящее изобретение не ограничивается конструкциями, в которых нетранслируемая область получена из 5'-нетранслируемой последовательности, которая сопутствует последовательности промотора. Лидерная последовательность также может быть получена из неродственной промоторной или кодирующей последовательности. Лидерные последовательности, подходящие в рамках настоящего изобретения, включают лидер Hsp70 кукурузы (US 5,362,865 и US 5,859,347) и омега элемент ВТМ.
Терминация транскрипции осуществляется 3'-нетранслируемой ДНК последовательностью, функционально связанной в векторе экспрессии с представляющей интерес молекулой РНК. 3'-нетранслируемая область рекомбинантной молекулы ДНК содержит сигнал полиаденилирования, который функционирует в растениях, вызывая присоединение аденилат-нуклеотидов к 3'-концу РНК. 3'-нетранслируемая область может быть получена из различных генов, которые экспрессируются в клетках растений. С этой целью обычно используют 3'-нетранслируемую область нопалинсинтазы, 3'-нетранслируемую область малой субъединицы гена Rubisco гороха, 3'-нетранслируемую область гена запасного белка 7S семян сои. Также подходят 3'-транскрибируемые нетранслируемые области, содержащие полиаденилат-сигнал генов опухолеиндуцирующей (Ti) плазмиды Agrobacterium.
Трансферные нуклеиновые кислоты
Трансферные нуклеиновые кислоты могут использоваться для доставки экзогенного полинуклеотида в клетку и включают одну, предпочтительно две бордерных последовательности и одну или больше представляющих интерес молекул РНК. Трансферная нуклеиновая кислота передачи может кодировать или может не кодировать селективный маркер. Предпочтительно трансферная нуклеиновая кислота является частью бинарного вектора в бактерии, где бинарный вектор дополнительно включает элементы, обеспечивающие репликацию вектора в бактерии, отбор или поддержание бактериальных клеток, содержащих бинарный вектор. После переноса в эукариотическую клетку трансферный компонент нуклеиновой кислоты бинарного вектора способен интегрироваться в геном эукариотической клетки или, в случае экспериментов с транзентной экспрессией, просто экспрессироваться в клетке.
При использовании в настоящем документе термин "внехромосомная трансферная нуклеиновая кислота" относится к молекуле нуклеиновой кислоты, которая может быть перенесена из бактерии, такой как бактерия Agrobacterium sp., в эукариотическую клетку, такую как клетка листа растения. Внехромосомная трансферная нуклеиновая кислота представляет собой генетический элемент, который хорошо известен как элемент, способный к переносу с последующей интеграцией нуклеотидной последовательности, содержащейся в его границах, в геном реципиентной клетки. В этом отношении трансферная нуклеиновая кислота фланкирована, как правило, двумя "бордерными" последовательностями, хотя в некоторых случаях может быть использован один бордер с одного конца, а второй конец перенесенной нуклеиновой кислоты образуется случайным образом в процессе переноса. Молекула РНК, представляющая интерес, обычно содержится между левой бордерной последовательностью и правой бордерной последовательностью трансферной нуклеиновой кислоты. Молекула РНК, содержащаяся в трансферной нуклеиновой кислоте, может быть функционально связана с рядом различных регуляторных элементов промоторов и терминаторов, которые способствуют ее экспрессии, то есть транскрипции и/или трансляции молекулы РНК. Трансферные ДНК (Т-ДНК) из таких видов Agrobacterium, как Agrobacterium tumefaciens или Agrobacterium rhizogenes, а также их искусственно созданные варианты/мутанты, вероятно, яляются наиболее полно описанными примерами трансферных нуклеиновых кислот. Другим примером является Р-ДНК ("растительная ДНК"), которая содержит подобные Т-ДНК-бордерам последовательности из растений.
При использовании в настоящем документе термин "Т-ДНК" относится, например, к Т-ДНК Ti-плазмиды Agrobacterium tumefaciens или из Ri-плазмиды Agrobacterium rhizogenes, или их вариантам, которые обеспечивают перенос Т-ДНК в клетки растений. Т-ДНК может содержать полную Т-ДНК, включающую правую и левую бордерные последовательности, но должна содержать лишь минимальные последовательности, требуемые в цис-конфигурации для переноса, то есть правую бордерную последовательность Т-ДНК. Т-ДНК настоящего изобретения содержат встроенную в них, в любом участке между правой и левой бордерными последовательностями (в случае присутствия), представляющую интерес молекулу РНК. Последовательности, кодирующие факторы, требуемые в транс-конфигурации для переноса Т-ДНК в клетку растения, такие как vir гены, могут быть встроены в Т-ДНК или могут присутствовать на том же репликоне, что и Т-ДНК, или предпочтительно находятся в транс-конфигурации на совместимом репликоне в хозяине Agrobacterium. Такие "бинарные векторные системы" хорошо известны в уровне техники. При использовании в настоящем документе "Р-ДНК" относится к трансферной нуклеиновой кислоте, выделенной из генома растения или человека, ее искусственно созданным вариантам/мутантам, и включает на каждом конце, или только на одном конце, последовательность, подобную T-ДНК бордеру.
При использовании в настоящем документе "бордерная" последовательность нуклеиновой кислоты может быть выделена из выбранного организма, такого как растение или бактерия, или может быть ее искусственно созданным вариантом/мутантом. Бордерная последовательность вызывает и облегчает перенос молекулы РНК, с которой она связана, и может облегчать собственную интеграцию в геном реципиентной клетки. В варианте осуществления бордерная последовательность имеет длину 10-80 пн. Бордерные последовательности из T-ДНК Agrobacterium sp. известны в уровне техники и включают последовательности, описанные в публикации Lacroix et al. (2008).
Хотя для переноса генов в клетки растений традиционно использовали только Agrobacterium sp., в настоящее время существует целый ряд систем, которые были идентифицированы/разработаны, которые действуют аналогично Agrobacterium sp. Недавно были генетически модифицированы несколько не относящихся к Agrobacterium видов, которые приобрели способность обеспечивать перенос генов (Chung et al., 2006; Broothaerts et al., 2005). Это включает Rhisobium sp. NGR234, Sinorhizobium meliloti и Mezorhizobium loti.
Прямой перенос эукариотических экспрессионных плазмид из бактерий в эукариотические хозяева был впервые осуществлен несколько десятилетий назад путем слияния клеток млекопитающих и протопластов плазмиданесущих Escherichia coli (Schaffher, 1980). С этого момента количество бактерий, способных доставлять гены в клетки млекопитающих, постоянно увеличивалось (Weiss, 2003), что было независимо установлено четырьмя группами исследователей (Sizemore at al., 1995; Courvalin et al., 1995; Powell et al., 1996; Darji et al., 1997).
При использовании в настоящем документе термины "трансфекция", "трансформация" и их вариации обычно используются попеременно. "Трансфицированные" или "трансформированные" клетки могут быть подвергнуты манипуляциям с целью введения представляющей интерес молекулы (молекул) РНК или могут быть дочерними клетками, полученными из них.
Рекомбинантные клетки
В изобретении также предложена рекомбинантная клетка, например, рекомбинантная клетка бактерии, клетка гриба, клетка растения, клетка насекомого или клетка животного, которая является клеткой-хозяином, трансформированная одной или более молекулами РНК или векторами, определенными в настоящем документе, или их комбинацией. Подходящие клетки настоящего изобретения включают любые клетки, которые могут быть трансформированы молекулой РНК или рекомбинантным вектором согласно настоящему изобретению. В примере трансформированная клетка-хозяин является мертвой.
Рекомбинантная клетка может быть клеткой в культуре, клеткой in vitro или в организме, таком как, например, растение, или в органе, таком как, например, семена или листья. Предпочтительно клетка находится в растении, более предпочтительно в семени растения. В одном варианте осуществления рекомбинантная клетка не является клеткой человека. Таким образом, в примере настоящее изобретение относится к организму, не относящемуся к человеку, включающему одну или более, или все молекулы РНК, раскрытые в настоящем документе.
В одном примере клетки являются клетками насекомого. В одном примере клетки насекомого получены из Trichoplusia.
Другим примером подходящей клетки-хозяина является электро-компетентная клетка HT115.
Клетки-хозяева, в которые вводят молекулу(ы) РНК, могут быть либо нетрансформированными клетками, либо клетками, которые уже трансформированы по меньшей мере одной нуклеиновой кислотой. Такие нуклеиновые кислоты могут участвовать в синтезе липидов или нет. Клетки-хозяева согласно настоящему изобретению могут быть способны эндогенно (то есть, естественно) экспрессировать молекулу(ы) РНК, определенную в настоящем документе, и в этом случае рекомбинантная клетка, полученная с ними, обладает повышенной способностью продуцировать молекулу(ы) РНК, или может быть способна продуцировать указанную молекулу(ы) РНК только после трансформации по меньшей мере одной молекулой РНК, определенной в настоящем документе. В одном примере клетка является клеткой, которую можно применять для продукции липида. В варианте осуществления рекомбинантная клетка согласно изобретению обладает повышенной способностью продуцировать неполярный липид, такой как TAG.
Клетки-хозяева согласно настоящему изобретению могут быть любыми клетками, способными экспрессировать по меньшей мере одну молекулу РНК, описанную в настоящем документе, и они включают клетки бактерий, грибов (включая дрожжи), паразитов, членистоногих, животных и растений. Примеры клеток-хозяев включают клетки Salmonella, Escherichia, Bacillus, Listeria, Saccharomyces, Spodoptera, Mycobacteria, Trichoplusia, Agrobacterium, клетки BHK (клетки почки новожденного хомяка), клетки MDCK, клетки CRFK, клетки CV-1, клетки COS (например, COS-7) и клетки Vero. Другими примерами клеток-хозяев являются E. coli, включая производные штаммы E. coli K-12; Salmonella typhi; Salmonella typhimurium, включая аттенуированные штаммы; Spodoptera frugiperda; Trichoplusia ni; и нетуморогенные мышиные миобластные клетки G8 (например, ATCC CRL 1246). Дополнительные подходящие клетки-хозяева млекопитающих включают другие линии клеток почек, другие линии клеток фибробластов (например, линии клеток эмбриональных фибробластов человека, мыши или курицы), линии клеток миеломы, клетки яичников китайского хомячка, клетки NIH/3T3 мыши, клетки LMTK и/или клетки HeLa.
В предпочтительном варианте осуществления клетка растения является клеткой семени, в частности, клеткой в семядоле или эндосперме семени. В одном варианте осуществления клетка является клеткой животного. Клетка животного может быть клеткой любого типа животного, такой как, например, клетка не относящегося к человеку животного, клетка не относящегося к человеку позвоночного, клетка не относящегося к человеку млекопитающего или клетками водных животных, таких как рыбы или ракообразные, беспозвоночные, насекомые и т.д. Примеры клеток водорослей, подходящих в качестве клеток-хозяев настоящего изобретения, включают, например, клетки Chlamydomonas sp. (например, Chlamydomonas reinhardtii), Dunaliella sp., Haematococcus sp., Chlorella sp., Thraustochytrium sp., Schizochytrium sp. и Volvox sp.
Трансгенные растения
В изобретении также предложено растение, включающее одну или более экзогенных молекул РНК, определенных в настоящем документе, клетку согласно настоящему изобретению, вектор согласно настоящему изобретению или их комбинацию. Термин "растение" относится к целым растениям, тогда как термин "его часть" относится к органам растения (например, листьям, стеблям, корням, цветкам, плодам), отдельным клеткам (например, пыльце), семени, частям семени, таким как зародыш, эндосперм, щиток или оболочка семени, тканям растения, таким как сосудистая ткань, клеткам растения и его потомству. При использовании в настоящем документе, части растения содержат клетки растения.
При использовании в настоящем документе термин "в растении", в отношени модификации растения, означает, что модификация произошла по меньшей мере в одной части растения, включая случай, когда модификация произошла во всем растении, и не исключая случай, когда модификация происходит только в одной или нескольких, но не всех, частях растения. Например, тканеспецифический промотор, как говорят, экспрессируется "в растении", даже несмотря на то, что он может экспрессироваться только в некоторых частях растения. Аналогичным образом, "полипептид фактора транскрипции, который повышает экспрессию одного или более гликолитических генов и/или генов биосинтеза жирных кислот в растении" означает, что повышение экспрессии происходит по меньшей мере в части растения.
При использовании в настоящем документе термин "растение" используется в самом широком смысле и включает любой организм в царстве Растений. Он также включает красные и бурые водоросли, а также зеленые водоросли. Он включает, без ограничения перечисленными, любые виды цветковых растений, трав, сельскохозяйственных или зерновых культур (например, масличные культуры, кукурузу, сою), кормовое или фуражное, плодовое или овощное растение, травянистое растение, древесное растение или дерево. Он не подразумевает ограничения растения какой-либо конкретной структурой. Он также относится к одноклеточному растению (например, микроводорослям). Термин "его часть" в отношении растения относится к растительной клетке и ее потомству, множеству растительных клеток, структуре, присутствующей на любой стадии развития растения, или ткани растения. Такие структуры включают, без ограничения перечисленными, листья, стебли, цветки, плоды, орехи, корни, семя, семенную оболочку, зародыши. Термин "растительная ткань" включает дифференцированные и недифференцированные ткани растений, включая те, которые присутствуют в листьях, стеблях, цветках, плодах, орехах, корнях, семенах, например, эмбриональной ткани, эндосперме, покровной ткани (например, эпидермисе, перидереме), сосудистой ткани (например, ксилеме, флоэме) или измельченной ткани (содержащей клетки паренхимы, колленхимы и/или склеренхимы), а также клетки в культуре (например, отдельные клетки, протопласты, каллус, эмбрионы и т.д.). Ткань растения может быть in planta, в культуре органов, культуре ткани или культуре клеток.
Различные количества жирных кислот 18:3 и 16:3 присутствуют в гликолипидах разных видов растений. Это используют, чтобы отличать растения 18:3, жирные кислоты которых с 3 двойными связями, как правило, всегда имеют цепи длиной С18 атомов, от растений 16:3, которые содержат как С16-, так и С18-жирные кислоты. В хлоропластах 18:3 активность ферментов, катализирующих превращение фосфатидата в диацилглицерин и диациглицерина в моногалактозилдиацилглицерин (МГД), значительно ниже, чем в хлоропластах 16:3. В листьях растений 18:3 хлоропласты синтезируют стеароил-АСР2 в строме, вводят первую двойную связь в насыщенную углеводородную цепь, а затем гидролизуют тиоэфир. Высвобождаемый олеат экспортируется через оболочки хлоропластов в мембраны эукариотической части клетки, вероятно, в эндоплазматический ретикулум, где он включается в ФХ. ФХ-связанные олеоильные группы в этих мембранах подвергаются десатурации и затем поступают обратно в хлоропласт. Связанные с МГД ацильные группы являются субстратами для введения третьей двойной связи с получением МГД с двумя линоленоильными остатками. Этот галактолипид характерен для растений 18:3, таких как, например, сложноцветные и бобовые. В фотосинтетически активных клетках растений 16:3, которые представлены, например, представителями семейств Apiaceae и Brassicaceae, два пути действуют параллельно, обеспечивая поступление МГД в тилакоиды. Объединенная "эукариотическая" последовательность в различной степени дополняется "прокариотическим" путем. Его ответы ограничиваются хлоропластом и приводят к типичному расположению ацильных групп, а также к их полной десатурации после их этерификации с образованием МГД. Прокариотические скелеты ДАГ несут С16:0 и продукты его десатурации в положении С-2, из которого удаляются С18: жирные кислоты. Положение C-1 занимают C18 жирные кислоты и в небольшой степени группы C16. Сходство в ДАГ-скелетах липидов сине-зеленых водорослей со скелетами, которые синтезируются в хлоропластном пути у растений 16:3, указывает на филогенетическую связь и оправдывает термин прокариот.
При использовании в настоящем документе термин "вегетативная ткань" или "вегетативная часть растения" является любой тканью, органом или частью растения кроме органов полового размножения растений. Органами полового размножения растений, в частности, являются семеноносные органы, цветки, пыльца, плоды и семена. Вегетативные ткани и части включают, по меньшей мере, листья растения, стебли (включая побеги и отростки, но исключая шаровидные стебли), клубни и корни, но исключая цветки, пыльцу, семена, в том числе семенную оболочку, зародыш и эндосперм, плоды, в том числе ткань мезокарпия, семеноносные коробочки и семеноносные головки. В одном варианте осуществления вегетативная часть растения является надземной частью растения. В другом или дополнительном варианте осуществления вегетативная часть растения является зеленой частью, такую как лист или стебель.
"Трансгенное растение" или его вариации относятся к растению, которое содержит трансген, не обнаруживаемый в растении дикого типа того же вида, разновидности или сорта. Трансгенные растения, как определено в рамках настоящего изобретения, включают растения и их потомство, которые были генетически модифицированы с применением рекомбинантных технологий, чтобы вызвать продукцию по меньшей мере одного полипептида, определенного в настоящем документе, в требуемом растении или его части. Части трансгенных растений имеют соответствующее значение.
Термины "семя" и "зерно" используются в настоящем документе попеременно. "Зерно" относится к зрелому зерну, такому как собранное зерно или зерно, которое все еще находится на растении, но готово к уборке, но может также относиться к зерну после набухания или прорастания, в зависимости от контекста. Зрелое зерно обычно имеет влажность меньше чем приблизительно 18%. В предпочтительном варианте осуществления содержание влаги в зерне находится на уровне, который обычно считается безопасным для хранения, предпочтительно от 5% до 15%, от 6% до 8%, от 8% до 10% или от 10% и 15%. При использовании в настоящем документе "развивающееся семя" относятся к семени до созревания, обычно обнаруживаемому в репродуктивных структурах растения после оплодотворения или прорастания, но также может относиться к таким семенам до созревания, которые выделены из растения. Зрелые семена обычно имеют влажность меньше чем приблизительно 12%.
При использовании в настоящем документе термин "запасающий орган растения" относится к части растения, приспособленной для хранения энергии, например, в форме белков, углеводов, липидов. Примерами запасающих органов растения являются семена, плоды, клубневидные корни и клубни. Предпочтительным запасающим органом растения согласно изобретению является семя.
При использовании в настоящем документе термин "фенотипически нормальный" относится к генетически модифицированному растению или его части, в частности, к запасающему органу, такому как семя, клубень или плод согласно изобретению, не обладающему существенно сниженной способностью к росту и размножению по сравнению с немодифицированным растением или его частью. Предпочтительно, биомасса, скорость роста, скорость прорастания, размер запасающего органа, размер семени и/или количество полученных жизнеспособных семян составляет не меньше 90% от соответствующего показателя растения, не содержащего указанный рекомбинантный полинуклеотид, при выращивании в идентичных условиях. Данный термин не охватывает признаки растения, которые могут отличаться от растения дикого типа, но которые не влияют на применимость растения в промышленных целях, такие как, например, фенотип "балерина" у листьев рассады. В варианте осуществления генетически модифицированное растение или его часть, которые являются фенотипически нормальными, включают рекомбинантный полинуклеотид, кодирующий сайленсирующий супрессор, функционально связанный с промотором, специфическим для запасающего органа растения, и имеет способность к росту или воспроизводству, которая по существу является такой же, как у соответствующего растения или его части, не включающих указанный полинуклеотид.
Растения, предложенные или предназначенные для применения при практическом осуществлении настоящего изобретения, включают однодольные и двудольные. В предпочтительных вариантах осуществления растения настоящего изобретения представляют собой культурные растения (например, злаковые и зернобобовые, кукурузу, пшеницу, картофель, тапиоку, рис, сорго, просо, маниок, ячмень или горох) или бобовые, такие как сою, бобы или горох. Растения могут быть выращены для производства съедобных корней, клубней, листьев, стеблей, цветков или плодов. Растения могут быть овощными растениями, вегетативные части которых употребляются в пищу. Растениями настоящего изобретения могут быть следующие: Acrocomia aculeate (пальма макауба), Arabidopsis thaliana, Aracinis hypogaea (арахис), Astrocaryum murumuru (мурумуру), Astrocaryum vulgare (тукума), Attalea geraensis (Indaia-rateiro), Attalea humilis (американская масличная пальма), Attalea oleifera (andaia), Attalea phalerata (урикури), Attalea speciosa (бабассу), Avena sativa (овес), Beta vulgaris (сахарная свекла), виды Brassica, такие как Brassica carinata, Brassica juncea, Brassica napobrassica, Brassica napus (канола), Camelina sativa (ложный лен), Cannabis sativa (конопля), Carthamus tinctorius (сафлор), Caryocar brasiliense (кариокар), Cocos nucifera (кокосовый орех), Crambe abyssinica (абиссинская капуста), Cucumis melo (дыня), Elaeis guineensis (африканская пальма), Glycine max (соя), Gossypium hirsutum (хлопок), виды Helianthus, такие как Helianthus annuus (подсолнечник), Hordeum vulgare (ячмень), Jatropha curcas (лекарственный орех), Joannesia princeps (ореховое дерево apapa), виды Lemna (ряска), такие как Lemna aequinoctialis, Lemna disperma, Lemna ecuadoriensis, Lemna gibba (ряска горбатая), Lemna japonica, Lemna minor, Lemna minuta, Lemna obscura, Lemna paucicostata, Lemna perpusilla, Lemna tenera, Lemna trisulca, Lemna turionifera, Lemna valdiviana, Lemna yungensis, Licania rigida (ойтикика), Linum usitatissimum (лен), Lupinus angustifolius (люпин), Mauritia flexuosa (пальма бурити), Maximiliana maripa (пальма инайа), виды Miscanthus, такие как Miscantus х giganteus и Miscanthus sinensis, виды Nicotiana (табак), такие как Nicotiana tabacum или Nicotiana benthamiana, Oenocarpus bacaba (bacaba-do-azeite), Oenocarpus bataua (pataua), Oenocarpus distichus (bacaba-de-leque), виды Oryza (рис), такие как Oryza sativa и Oryza glaberrima, Panicum virgatum (просо прутьевидное), Paraqueiba paraensis (умари), Persea amencana (авокадо), Pongamia pinnata (индийский бук), Populus trichocarpa, Ricinus communis (клещевина обыкновенная), виды Saccharum (сахарный тростник), Sesamum indicum (кунжут), Solanum tuberosum (картофель), виды Sorghum, такие как Sorghum bicolor, Sorghum vulgare, Theobroma grandiforum (купуасу), виды Trifolium, Trithrinax brasiliensis (бразильская игольчатая пальма), виды Triticum (пшеница), такие как Triticum aestivum, Zea mays (кукуруза), люцерна (Medicago sativa), рожь (Secale cerale), батат (Ipomoea batatus), маниок (Manihot esculenta), кофе (виды Cofea), ананас (Anana comosus), цитрусовые деревья (виды Citrus), какао (Theobroma cacao), чай (Camellia senensis), банан (виды Musa), авокадо (Persea Americana), инжир (Ficus casica), гуава (Psidium guajava), манго (Magnifer indica), олива европейская (Olea europaea), папайя (Carica papaya), кешью (Anacardium occidentale), макадамия (Macadamia intergrifolia) и миндаль (Prunus amygdalus). Например, растениями согласно изобретению может быть Nicotiana benthamiana.
Другие предпочтительные растения включают травы C4 такой как, в дополнение к указанным выше, Andropogon gerardi, Bouteloua curtipendula, B. gracilis, Buchloe dactyloides, Schizachyrium scoparium, Sorghastrum nutans, Sporobolus cryptandrus; травы C3, такие как Elymus canadensis, бобовые Lespedeza capitata и Petalostemum villosum, полукустарник Aster azureus; и древесные растения, такие как Quercus ellipsoidalis и Q. macrocarpa. Другие предпочтительные растения включают травы C3.
В предпочтительном варианте осуществления растение является покрытосемянным растением.
В варианте осуществления растение является масличным растением, предпочтительно масличным культурным растением. При использовании в настоящем документе "масличное растение" является видом растения, используемым для промышленного производства липида из семян растения. Масличное растение может быть, например, масличным рапсом (таким как канола), кукурузой, подсолнечником, сафлором, соей, сорго, льном (льном обыкновенным) или сахарной свеклой. Кроме того, масличное растение может быть другим растением Brassica, хлопком, арахисом, маком, брюквой, горчицей, клещевиной, кунжутом, сафлором, Jatropha curcas или ореховым растением. Растение может вырабатывать высокие уровни липидов в плодах, как олива, масличная пальма или кокос. Плодовоовощными растениями, к которым может быть применено настоящее изобретение, являются салат-латук, цикорный салат или овощные культуры Brassica, включая капусту, брокколи или цветную капусту. Настоящее изобретение может применяться в отношении табака, тыквы, моркови, клубники, томатов или перца.
В предпочтительном варианте осуществления трансгенное растение является гомозиготным по каждому гену, который был введен (трансгену), чтобы его потомство не расщеплялось на требуемый фенотип. Трансгенное растение также может быть гетерозиготным по введенному трансгену(ам), предпочтительно однородно гетерозиготным по трансгену, таким как, например, в потомстве F1, которое были выращено из гибридного семени. Такие растения могут обеспечивать преимущества, такие как гибридная сила, известная в уровне техники.
Трансформация
Молекулы РНК, раскрытые в настоящем документе, могут быть стабильно введены в вышеуказанные клетки-хозяева и/или не относящиеся к человеку организмы, такие как растения. Во избежание неопределенности пример настоящего описания охватывает вышеуказанное растение, стабильно трансформированное молекулой РНК, раскрытой в настоящем документе. При использовании в настоящем документе термины "стабильная трансформация", "стабильно трансформированный" и их вариации относятся к интеграции молекулы РНК или нуклеиновой кислоты, кодирующей ее, в геном клетки, в результате чего они передаются клеткам потомства во время деления клеток без необходимости в положительном отборе на их присутствие. Стабильные транформанты или их потомство могут быть идентифицированы любым способом, известных в уровне техники, таким как Саузерн-блоттинг на хромосомной ДНК или гибридизация in situ геномной ДНК, которые позволяют производить их отбор.
Трансгенные растения могут быть получены с помощью методов, известных в уровне техники, таких как методы, описанные в общем виде в Slater et al., Plant Biotechnology - The Genetic Manipulation of Plants, Oxford University Press (2003), и Christou and Klee, Handbook of Plant Biotechnology, John Wiley and Sons (2004).
В варианте осуществления растения могут быть трансформированы путем нанесения молекулы РНК согласно настоящему раскрытию на поверхность растения или его части. Например, молекула РНК может быть представлена в виде состава с подходящим носителем и распылена, напылена или иным образом нанесена на поверхность растения или его части. Таким образом, в примере способы настоящего изобретения охватывают введение молекулы РНК, раскрытой в настоящем документе, в растение, где способ включает нанесение композиции, содержащей молекулу РНК, на поверхность растения или его части.
Агробактериальный перенос представляет собой широко применяемую систему введения генов в клетки растений, поскольку ДНК можно вводить в клетки всего растения, ткани, органы растения или эксплантаты в культуре ткани либо для их транзиентной экспрессии, либо для стабильной интеграции ДНК в геном клетки растения. Например, могут использоваться методы с погружением цветков (in planta). Применение агробактериальных растительных интеграционных векторов для введения ДНК в клетки растений известно в уровне техники. Область ДНК, которая будет перенесена, определяется бордерными последовательностями, а расположенная межу ними ДНК (T-ДНК) обычно встраивается в геном растения. Этот метод предпочтителен из-за удобной и определенной природы переноса генов.
Методы ускорения, которые могут использоваться, включают, например, бомбардировку микрочастицами и т.п. Одним примером способа доставки трансформирующих молекул нуклеиновой кислоты в растительные клетки является бомбардировка микрочастицами. Этот метод был описан в обзоре Yang et al., Particle Bombardment Technology for Gene Transfer, Oxford Press, Oxford, England (1994). Небиологические частицы (микрочастицы) могут быть покрыты нуклеиновыми кислотами и доставлены в клетки, например, незрелые зародыши, под действием толкающей силы. Примеры частиц включают частицы из вольфрама, золота, платины и т.п.
В другом способе могут быть стабильно трансформированы пластиды. Способы, раскрытые для трансформации пластид в высших растениях, включают баллистическую доставку частиц с ДНК, содержащей селективный маркер и направление ДНК в геном пластид путем гомологичной рекомбинации (US 5,451,513, US 5,545,818, US 5,877,402, US 5,932479 и WO 99/05265). Другие методы трансформации клеток также могут использоваться и включают, без ограничения перечисленными, введение ДНК в растения путем прямого переноса ДНК в пыльцу, прямой инъекции ДНК в репродуктивные органы растения или прямой инъекции ДНК в клетки незрелых зародышей, с последующей регидратацией высушенных зародышей.
Регенерация, развитие и культивирование растений из одиночных трансформантов протопластов растения или из различных трансформированных эксплантатов хорошо известны в данной области техники (Weissbach et al., в публикации: Methods for Plant Molecular Biology, Academic Press, San Diego, Calif. (1988)). Этот процесс регенерации и выращивания обычно включает этапы отбора трансформированных клеток, выращивания этих обособленных клеток на обычных стадиях развития зародыша до стадии укоренившегося проростка. Трансгенные зародыши и семена регенерируют одинаково. Полученные трансгенные укоренившиеся проростки затем выращивают в подходящей среде для выращивания растений, такой как грунт.
Развитие или регенерация растений, содержащих чужеродный, экзогенный ген, хорошо известны в уровне техники. Предпочтительно, регенерированные растения являются самоопыляемыми с получением гомозиготных трансгенных растений. Или же пыльцу, полученную из регенерированных растений, скрещивают с выращенными из семян растениями агрономически важных линий. И наоборот, пыльцу из растений этих важных линий используют для опыления регенерированных растений. Трансгенное растение настоящего изобретения, содержащее требуемый полинуклеотид, выращивают с применением способов, хорошо известных специалистам в данной области.
Для подтверждения присутствия трансгенов в трансгенных клетках и растениях может быть проведена амплификация методом полимеразной цепной реакции (ПЦР) или Саузерн-блот анализ с применением способов, известных специалистам в данной области. Продукты экспрессии трансгенов могут быть обнаружены любым из различных способов, в зависимости от природы продукта, которые включают Нозерн-блот гибридизацию, Вестерн-блоттинг и ферментный анализ. После получения трансгенных растений они могут быть выращены с получением тканей или частей растения, обладающих требуемым фенотипом. Ткань растения или части растения могут быть собраны, и/или собраны семена. Семена могут служить в качестве источника для выращивания дополнительных растений с тканями или частями, обладающими нужными характеристиками. Предпочтительно, вегетативные части растения собирают в тот момент, когда выход неполярных липидов является наиболее высоким. В одном варианте осуществления вегетативные части растения собирают примерно во время цветения или после начала цветения. Предпочтительно части растения собирают приблизительно во время начала старения, обычно проявляющегося пожелтением и высыханием листьев.
Трансгенные растения, полученные с помощью агробактериальных или других способов трансформации, обычно содержат один генетический локус на одной хромосоме. Такие трансгенные растения могут именоваться гемизиготными по добавленному гену(ам). Более предпочтительным является трансгенное растение, которое является гомозиготным по добавленному гену(ам), то есть трансгенное растение, которое содержит два добавленных гена, один ген в одном и том же локусе на каждой хромосоме в хромосомной паре. Гомозиготное трансгенное растение может быть получено при самооплодотворении гемизиготоного трансгенного растения, проращивании некоторых полученных семян и анализе полученных растений на присутствие гена, представляющего интерес.
Следует также понимать, что два разных трансгенных растения, которые содержат два независимо расщепляющихся экзогенных гена или локуса, также могут скрещиваться (сочетаться) с получением потомства, которое содержит оба набора генов или локусов. Самоопыление соответствующего потомства F1 может давать растения, которые являются гомозиготными по обоим экзогенным генам или локусам. Также предусмотрено обратное скрещивание с родительским растением и ауткроссинг с нетрансгенным растением, как и вегетативное размножение. Аналогичным образом, трансгенное растение может быть скрещено со вторым растением, включающим генетическую модификацию, такую как мутантный ген, после чего идентифицируют потомство, содержащее и трансген, и генетическую модификацию. Описания других способов скрещивания, которые обычно используются для различных признаков и культур, можно найти в публикации Fehr, In: Breeding Methods for Cultivar Development, Wilcox J. ed., American Society of Agronomy, Madison Wis. (1987).
Композиции
Молекулы РНК согласно настоящему изобретению могут быть предоставлены в виде различных композиций. Например, молекулы РНК могут быть в твердой форме, в форме мази, геля, крема, порошка, пасты, суспензии, коллоида, пены или аэрозоля. Твердые формы могут включать дусты, порошки, гранулы, пеллеты, пилюли, пастилки, таблетки, наполненные пленки (включая покрытия семян) и т.п., которые могут быть воднодиспергируемыми ("смачиваемыми"). В одном примере композиция находится в форме концентрата.
В примере молекулы РНК могут быть предоставлены в виде композиции для нанесения. В примере композиция стабилизирует молекулу РНК в композиции и/или in vivo. Например, молекулы РНК могут быть предоставлены в липидной композиции. Например, молекулы РНК могут быть предоставлены в липосомах. В примере композиция включает средство, способствующее трансфекции.
В примере молекулы РНК могут быть включены в композиции, подходящие для применения на поле. В примере поле включает растения. Подходящие растения включают культурные растения (например, злаковые и зернобобовые, кукурузу, пшеницу, картофель, тапиоку, рис, сорго, сою, просо, маниок, ячмень или горох) или бобовые. Растения могут быть выращены для производства съедобных корней, клубней, листьев, стеблей, цветков или плодов. В примере культурное растение является злаковым растением. Примеры злаковых растений включают, без ограничения перечисленными, пшеницу, ячмень, овес, сорго и рожь. В этих примерах молекула РНК может быть включена в композицию для введения в растение или любую часть растения любым подходящим способом. Например, композиция может быть приготовлена для введения в листья, стебель, корни, плоды, овощи, зерна и/или бобы растения. В одном примере молекула РНК включена в композицию для введения в листья растения и может распыляться на листья растения.
В зависимости от требуемой композиции молекулы РНК, описанные в настоящем документе, могут быть включены в композицию с множеством других веществ. Примеры веществ включают одно или более суспендирующих веществ, гранулирующих веществ, основ, буферов, веществ, придающих горький вкус, ароматизаторов, консервантов, пропеллентов, тиксотропных веществ, добавок, предохраняющих от замерзания, и красителей.
В других примерах, композиции молекулы РНК могут включать инсектицид, пестицид, фунгицид, антибиотик, репеллент от насекомых, противопаразитное средство, противовирусное средство или нематоцид.
В другом примере молекулы РНК могут быть включены в фармацевтические композиции. Такие композиции, как правило, включают молекулу РНК, описанную в настоящем документе, и фармацевтически приемлемый носитель. При использовании в настоящем документе фраза "фармацевтически приемлемый носитель" включает растворители, дисперсионные среды, покрытия, противобактериальные и противогрибковые средства, изотонические вещества и вещества, задерживающие абсорбцию, и т.п., совместимые с фармацевтическим применением. В композиции также могут быть включены дополнительные активные соединения.
Фармацевтическую композицию изготавливают совместимой с ее предполагаемым путем введения. Примеры путей введения включают парентеральное, например, внутривенное, внутрикожное, подкожное, ингаляционное, трансдермальное (наружный), чресслизистое, пероральное и ректальное введение.
В одном варианте осуществления активные соединения приготовлены с носителями, которые предохраняют соединение от быстрого выведения из организма, например, в композиции с контролируемым высвобождением, включая имплантаты и микроинкапсулированные системы доставки. Могут использоваться биоразлагаемые, биосовместимые полимеры, такие как этилен-винилацетат, полиангидриды, полигликолевая кислота, коллаген, полиортоэфиры и полимолочная кислота. Например, в качестве фармацевтически приемлемых носителей также могут использоваться липосомные суспензии. Они могут быть изготовлены согласно способам, известным специалистам в данной области, например, как описано в US 4,522,811.
Молекулы РНК согласно настоящему изобретению могут быть предоставлены в наборе или упаковке. Например, молекулы РНК, раскрытые в настоящем документе, могут быть упакованы в подходящем контейнере с письменными инструкциями по получению вышеуказанной клетки или организма, или лечению заболевания.
Способы контроля не относящихся к человеку организмов
В примере молекулы РНК согласно настоящему изобретению могут применяться для котроля численности не относящихся к человеку организмов, таких как насекомые. Такие применения включают введение молекулы РНК согласно настоящему изобретению с помощью различных способов. В примере молекулы РНК согласно настоящему изобретению могут быть предоставлены насекомым в виде приманки для поедания насекомыми. В другом примере молекулы РНК могут быть распылены на насекомых, как требуется. В другом примере молекулы РНК могут быть распылены на растение или сельскохозяйственную культуру для защиты указанного растения или сельскохозяйственной культуры от насекомых. Примеры сельскохозяйственных культур включают хлопок, кукурузу, томат, нут, голубиный горох, люцерну, рис, сорго и коровий горох.
В примере молекулы РНК могут быть предоставлены для изменения поведения насекомого. В другом примере молекулы РНК могут быть предоставлены для уничтожения насекомых. В другом примере молекулы РНК могут быть предоставлены для уменьшения плодуктивности насекомого. Примеры насекомых-мишеней включают домовых насекомых. Другие примерные насекомые-мишени включают насекомых, питающихся растительными соками, таких как тли (например, Myzus persicae, Metopolophium dirhodum, Rhopalosiphum padi, Aphis glycines, Aphis fabae). Другие примерные насекомые-мишени включают паукообразных, комаров, эктопаразитов, мух, паутинных клещей, трипсов, клещей, куриных клещей, муравьев, тараканов, термитов, сверчков, включая домовых сверчков, чешуйниц, сеноедов, жуков, уховерток, москитов и блох. Другие примерные насекомые-мишени включают сельскохозяйственных вредителей. Примеры включают вредителей, питающихся соками растений, таких как клопы и тли, грызущих насекомых, таких как гусеницы, жуки, черви, трущие насекомые, такие как трипсы и слизни, моль, мухи, такие как плодовые мушки, вредители зерна, такие как зерновой точильщик, долгоносики и зерновая моль.
В варианте осуществления насекомое притается соком растения. В этом примере молекула РНК может обладать антисмысловой активностью в отношении MpC002 и/или MpRack-1. В варианте осуществления насекомое, притающееся соком растения, является тлей. В другом варианте осуществления тлей является Myzus persicae.
В варианте осуществления насекомое-мишень является муравьем (например, Linepithema humile), хлопковой совкой (Helicoverpa armigera) или мясной мухой (например, Lucilia cuprina). В варианте осуществления насекомым-мишенью является Helicoverpa armigera, и молекула РНК обладает антисмысловой активностью в отношении гена ABC-переносчика white (ABC white). В другом варианте осуществления насекомым-мишенью является Linepithema humile, и молекула РНК обладает антисмысловой активностью в отношении нейропептида активации биосинтеза феромонов (PBAN). В другом варианте осуществления целевым насекомым является Lucilia cuprina, и молекула РНК обладает антисмысловой активностью в отношении одного или более генов, кодирующих белки, выбранные из группы, состоящей из каталитической субъединицы A протонной АТФ-азы V-типа, РНКазы 1/2, хитинсинтазы, рецептора экдизона и гамма-тубулин 1/1-подобного.
В вышеуказанном варианте осуществления композиции и молекулы РНК, раскрытые в настоящем документе, могут быть предоставлены в дозирующем устройстве. В примере дозирующее устройство является ловушкой или приманкой. В варианте осуществления ловушка и/или приманка включает съедобную приманку, включающую молекулу(ы) РНК, раскрытую в настоящем документе.
В варианте осуществления настоящее изобретение охватывает способы контроля поведения насекомых, где способ включает распыление, опыление или иное нанесение молекул РНК, раскрытых в настоящем документе, на насекомых. В этом варианте осуществления молекулы РНК могут быть распылены, напылены или иным образом нанесены непосредственно на насекомых. В другом варианте осуществления молекулы РНК могут быть распылены, напылены или иным образом нанесены на растения или сельскохозяйственные культуры до заражения насекомыми.
В одном варианте осуществления изобретения насекомое или паукообразное могут относиться к следующим отрядам: Acari, Arachnida, Anoplura, Blattodea, Coleoptera, Collembola, Dermaptera, Dictyoptera, Diplura, Diptera, Embioptera, Ephemeroptera, Grylloblatodea, Hemiptera, Heteroptera, Homoptera, Hymenoptera, Isoptera, Lepidoptera, Mallophaga, Mecoptera, Neuroptera, Odonata, Orthoptera, Phasmida, Phithiraptera, Plecoptera, Protura, Psocoptera, Siphonaptera, Siphunculata, Thysanura, Sternorrhyncha, Strepsiptera, Thysanoptera, Trichoptera, Zoraptera и Zygentoma.
В предпочтительных, но не ограничивающих, вариантах осуществления изобретения насекомое или паукообразное выбрано из группы, состоящей из следующего: (1) Acari: клещи, включая Ixodida (клещи) (2) Arachnida: Araneae (пауки) и Opiliones (сенокосцы), примеры: Latrodectus mactans (черная вдова) и Loxosceles recluse (коричневый паук-отшельник) (3) Anoplura: вши, такие как Pediculus humanus (человеческая вошь) (4) Blattodea: тараканы, в том числе рыжий таракан (Blatella germanica), рода Periplaneta, в том числе американский таракан (Periplaneta americana) и австралийский таракан (Periplaneta australiasiae), рода Blatta, в том числе восточный таракан (Blatta orientalis) и рода Supella, включая коричневополосого таракана (Supella longipalpa). Наиболее предпочтительной мишенью является рыжий таракан (Blatella germanica). (5) Coleoptera: жуки, примеры включают в себя: семейство бострихоидных (семейство Bostrichoidea); Dendroctonus spp. (лубоед, южный сосновый жук, короед); кожееды (Anthrenus spp, Attagenus spp); усач домовый (семейство Cerambycidae: Hylotrupes bajulus); Anobium punctatum; Tribolium spp (мучной жук); Trogoderma granarium (кожеед зерновой); Oryzaephilus sarinamensis (суринамский рисоед) и т.д. (книжная моль) (6) Dermaptera: семейство уховерток (7) Diptera: комары (Culicidae) и мухи (Brachycera), примеры: Anophelinae, такие как Anopheles spp. и Culicinae, такие как Aedes fulvus; Tabanidae, такие как Tabanus punctifer (западный слепень), Glossina morsitans morsitans (муха цеце), бабочницы (Psychodidae) и Calyptratae, такие как Musca domestica (комнатная муха), мухи (семейство Sarcophagidae) и т.д. (8) Heteroptera: клопы, такие как Cimex lectularius (постельный клоп) (9) Hymenoptera: осы (Apocrita), включая муравьев (Formicoidea), пчел (Apoidea): Solenopsis invicta (красный огненный муравей), Monomorium pharaonis (фараонов муравей), Camponotus spp (муравьи-плотники), Iasius niger (черный садовый муравей), Tetramorium caespitum (дерновый муравей), Myrmica rubra (красный муравей), Formica spp (древесные муравьи), Crematogaster lineolata (муравей-акробат), Iridomyrmex humilis (аргентинский муравей), Pheidole spp. (Крупноголовые муравьи), Dasymutilla occidentalis (бархатный муравей) и т.д. (10) Isoptera: термиты, примеры включают: Amitermes floridensis (флоридский подземный темнокрылый термит), восточный подземный термит (Reticulitermes flavipes), R. hesperus (западный подземный термит), Coptotermes formosanus (тайваньский подземный термит), Incisitermes minor (западный древесный термит), Neotermes connexus (лесной термит) и Termitidae (11) Lepidoptera: моль, примеры включают: Tineidae и Oecophoridae, например, Tineola bisselliella (платяную моль) и Pyralidae, такие как Pyralis farinalis (огневку мучную) и т.д. (12) Psocoptera: сеноеды (13) Siphonaptera: блохи, такие как Pulex allerans (14) Sternorrhyncha: тля (Aphididae) (15) Zygentoma: чешуйницы, примеры: Thermobia domestica и Lepisma saccharina.
Другие целевые насекомые или паукообразные включают домовых насекомых, эктопаразитов и насекомых и/или паукообразных, важных в отношении здравоохранения и гигиены, таких как, в качестве примера, но не ограничения, мухи, паутинные клещи, трипсы, клещи, куриный клещ, муравьи (например, при направленном воздействии на PBAN), тараканы, термиты, сверчки, включая домовых сверчков, чешуйницы, сеноеды, жуки, уховертки, комары и блохи. Более предпочтительными мишенями являются тараканы (Blattodea), такие как Blatella spp. (например, Blatella germanica (рыжий таракан)), Periplaneta spp. (например, Periplaneta americana (американский таракан) и Periplaneta australiasiae (австралийский таракан)), Blatta spp. (например, Blatta orientalis (восточный таракан)) и Supella spp. (например, Supella longipalpa (коричневополосый таракан); муравьи (Formicoidea), такие как, без ограничения перечисленными, виды Solenopsis (например, Solenopsis invicta (красный огненный муравей)), виды Monomorium (например, Monomorium pharaonis (фараонов муравей)), виды Camponotus (например, Camponotus spp (муравьи-плотники)), виды Lasius (например, Lasius niger (черный садовый муравей)), виды Tetramorium (например, Tetramorium caespitum (дерновый муравей)), виды Myrmica (например, Myrmica rubra (красный муравей)), Formica spp (древесные муравьи), Crematogaster spp. (например, Crematogaster lineolata (муравей-акробат)), Iridomyrmex spp. (например, Iridomyrmex humilis (аргентинский муравей)), Pheidole spp. (крупноголовые муравьи) и Dasymutilla spp. (например, Dasymutilla occidentalis (бархатный муравей)), термиты (Isoptera и/или Termitidae), такие как, без ограничения перечисленными, виды Amitermes (например, Amitermes floridensis (флоридский подземный темнокрылый термит)), виды Reticulitermes (например, Reticulitermes flavipes (восточный подземный термит), Reticulitermes hesperus (западный подземный термит)), виды Coptotermes (например, Coptotermes formosanus (тайваньский подземный термит), Incisitermes spp. (например, Incisitermes minor (западный древесный термит)), Neotermes spp. (например, Neotermes connexus (лесной термит)).
В варианте осуществления целевая РНК кодирует ацетолактатсинтазу насекомого.
Молекулы РНК согласно изобретению при доставке и/или экспрессии в растении могут обладать широким спектром желательных свойств, которые влияют, например, на агрономический признак, устойчивость к насекомым (например, в результате направленного воздействия на такие гены, как MpC002, MpRack-1 и ген ABC-переносчика), устойчивость к болезням (например, в результате направленного воздействия на такие гены, как LanR), устойчивость к гербицидам, стерильность, характеристики зерна и т.п. Молекула РНК-мишени может участвовать в метаболизме масла, крахмала, углеводов, питательных веществ и т.д., или может отвечать за синтез белков, пептидов, жирных кислот, липидов, частоту рекомбинации (в результате направленного воздействия на такие гены, как DDM1 и FANCM), восков, масел (в результате направленного воздействия на такие гены, как TOR), крахмалов, сахаров, углеводов, вкусовых и ароматических веществ, токсинов, каротиноидов, гормонов (в результате направленного воздействия на такие гены, как EIN2, NCED1 и NCED2), полимеров, флавоноидов (в результате направленного воздействия на такой ген как халконсинтаза), запасных белков, фенольных кислот, алкалоидов, лигнинов, дубильных веществ, целлюлоз, гликопротеинов, гликолипидов и др.
В конкретном примере растения вырабатывают повышенные уровни ферментов для продукции масел в растениях, таких как Капустные, например, рапс или подсолнечник, сафлор, лен, хлопок, соя или кукуруза; ферментов, участвующих в синтезе крахмала в растениях, таких как картофель, кукуруза и злаки, такие как пшеница, ячмень или рис; ферментов, которые синтезируют, или белков, которые сами представляют собой, природные лекарственные средства, такие как фармацевтические или ветеринарные продукты.
В другом варианте осуществления молекула РНК согласно настоящему изобретению направлена на профилактическое или терапевтическое лечение инфекции, вызванной грибковым патогеном, выбранным из группы, состоящей из следующих: Altemaria spp.; Armillaria mellae; Arthrobotrys oligosporus; Blumeria graminis (в результате направленного воздействия на гены Mlo с применением молекулы РНК, описанной в Примере 17), Boletus granulatus; Botritis cinerea; Botrytis fabae; Candida albicans; Claviceps purpurea; Cronartium ribicola; Epicoccum purpurescens; Epidermophyton floccosum; Fomes annosus; Fusarium oxysporum; Gaeumannomyces graminis var. tritici; Glomerella cingulata; Gymnosporangium juniperi-virginianae; Microsporum canis; Monilinia fructicola; Physoderma alfalfae; Phytopthera infestans; Pityrosporum orbiculare (Malassezia furfur); Polyporus sulphureus; Puccinia spp.; Saccharomyces cerevisiae; Septoria apiicola; Trichophyton rubrum; T. mentagrophytes; Ustilago spp .; Venturia inaequalis; и Verticillium dahliae.
Примеры состояний, подлежащих лечению
Молекулы РНК согласно настоящему изобретению могут применяться в способах различных состояний. В некоторых примерах настоящее изобретение относится к способу лечения рака, включающему введение молекулы РНК, раскрытой в настоящем документе. Термин "рак" относится или описывает физиологическое состояние у млекопитающих, которое обычно характеризуется нерегулируемым ростом/пролиферацией клеток. Примеры рака включают, без ограничения перечисленным, карциному, лимфому, бластому, саркому и лейкоз или лимфоидные злокачественные новообразования. Более конкретные примеры таких форм рака включают, без ограничения перечисленными, плоскоклеточный рак (например, эпителиальный плоскоклеточный рак), рак легкого, включая мелкоклеточный рак легкого, немелкоклеточный рак легкого, аденокарциному легкого и плоскоклеточную карциному легкого, рак брюшины, гепатоцеллюлярный рак, рак желудка, включая рак желудочно-кишечного тракта и стромальный рак желудочно-кишечного тракта, рак поджелудочной железы, глиобластому, рак шейки матки, рак яичника, рак печени, рак мочевого пузыря, рак мочевыводящих путей, гепатому, рак молочной железы, рак толстой кишки, рак прямой кишки, рак толстой и прямой кишки, рак эндометрия или матки, карциному слюнной железы, рак почки, рак предстательной железы, рак вульвы, рак щитовидной железы, карциному печени, карциному анального канала, карциному полового члена, меланому, поверхностную распространяющуюся меланому, злокачественную лентиго-меланому, акральную лентигинозную меланому, узловую меланому, множественную миелому и B-клеточную лимфому (включая низкодифференцированную/фолликулярную неходжкинскую лимфому (НХЛ); лимфому из мантийных клеток; СПИД-ассоциированную лимфому; и макроглобулинемию Вальденстрема); хронический лимфолейкоз (ХЛЛ); острый лимфобластный лейкоз (ОЛЛ); волосатоклеточный лейкоз; хронический миелобластный лейкоз; и посттрансплантационное лимфопролиферативное нарушение (PTLD), а также аномальную пролиферацию сосудов, связанную с факоматозами, отеками (такими как отеки, ассоциированные с опухолями головного мозга), синдром Мейгса, раком головного мозга, а также рак головы и шеи, и ассоциированные метастазы. Таким образом, в примере настоящее изобретение относится к способу лечения рака молочной железы, яичников, толстой кишки, предстательной железы, легкого, мозга, головного кожи, печени, желудка, поджелудочной железы или гемобластоза.
В других примерах способ, описанный в настоящем документе, применяется для лечения онкологических заболеваний, связанных с мутациями в генах BRCA1, BRCA2, PALB2 или RAD51B, RAD51C, RAD51D или родственных генах. В других примерах способ, описанный в настоящем документе, применяется для лечения онкологических заболеваний, связанных с мутациями в генах, связанными с репарацией неспаренных оснований ДНК, таких как MSH2, MLH1, PMS2, и родственных генах. В других примерах способ, описанный в настоящем документе, применяется для лечения онкологических заболеваний с сайленсингом генов репарации ДНК, таких как BRCA1, MLH1 или RAD51B, RAD51C, или RAD51D.
В других примерах изобретения способ, описанный в настоящем документе, применяется для уничтожения клеток с нарушенными процессами репарации ДНК. Например, клетки со сниженной репарацией ДНК могут аберрантно экспрессировать ген, участвующий в репарации ДНК, синтезе ДНК или гомологичной рекомбинации. Примеры генов включают: XRCC1, ADPRT (PARP-1), ADPRTL2, (PARP-2), БЕТА-ПОЛИМЕРАЗУ, CTPS, MLH1, MSH2, FANCD2, PMS2, p53, p21, PTEN, RPA, RPAl, RPA2, RPA3, XPD, ERCC1, XPF, MMS19, RAD51, RAD51B, RAD51C, RAD51D, DMC1, XRCCR, XRCC3, BRCA1, BRCA2, PALB2, RAD52, RAD54, RAD50, MREU, NB51, WRN, BLM, KU70, KU80, банкомат, ATR CPIK1, CHK2, FANCA, FANCB, FANCC, FANCD1, FANCD2, FANCE, FANCF, FANCG, FANCC, FANCD1, FANCD2, FANCE, FANCF, FANCG, RAD1 и RAD9. В примере способ, описанный в настоящем документе, применяется для уничтожения клеток с мутантным геном-супрессором опухоли. Например, клетки могут иметь одну или более мутаций в BRCAl или BRCA2.
В других примерах изобретения способ, описанный в настоящем документе, применяется для лечения трансформированных вирусом клеток. В других примерах изобретения способ, описанный в настоящем документе, применяется для уничтожения клеток, трансформированных латентным вирусом. Примеры латентных вирусов включают ЦМВ, ВЭБ, вирус простого герпеса (1 и 2 типа) и вирус ветряной оспы. В других примерах изобретения способ, описанный в настоящем документе, применяется для лечения активных вирусных инфекций, вызванных вирусами, которые приводят к раку, иммуннодефициту, гепатиту, энцефалиту, пневмониту или респираторным заболеваниям. Примеры вирусов включают парвовирус, поксвирус, вирус герпеса.
В других примерах изобретения способ, описанный в настоящем документе, применяется для лечения вируса Зика, колорадской клещевой лихорадки (вызванной колтивирусом, РНК-вирусом), лихорадки Западного Нила (энцефалита, вызванного флавивирусом, который в основном встречается на Ближнем Востоке и в Африке). Желтой лихорадки, бешенства (вызванного рядом разных штаммов нейротропных вирусов семейства Rhabdoviridae), вирусного гепатита, гастроэнтерита (вирусного) - острого вирусного гастроэнтерита, вызванного вирусами Норуолк и норуолк-подобными вирусами, ротавирусов, калицивирусов и астровирусов, полиомиелита, гриппа, вызванного ортомиксовирусами, которые могут подвергаться частым антигенным вариациям, кори, парамиксовирусов, эпидемического паротита, респираторных синдромов, включая вирусную пневмонию и острые респираторные синдромы, включая круп, вызванные различными вирусами, которые в совокупности называются острыми респираторными вирусами, и респираторных заболеваний, вызванных респираторно-синцитиальным вирусом.
ПРИМЕРЫ
Пример 1. Материалы и методы
Синтез генетических конструкций
Для конструирования типичной конструкции кпРНК была идентифицирована область РНК-мишени длиной примерно 100-1000 нуклеотидов, обычно 400-600 нуклеотидов. В одном примере 5'-половина последовательности и приблизительно 130 нт фланкирующей области и, аналогично, 3'-половина и 130 нт фланкирующей области были ориентированы в антисмысловой ориентации относительно промотора. Эти последовательности прерывали нуклеотидной смысловой последовательностью 400-600 (Фигура 1А). Перед 5'-концом полученной конструкции помещали промотор, такой как промотор РНК-полимеразы T7 или SP6, а 3'-конец включал сайт рестрикции, чтобы обеспечить терминацию транскрипции in vitro.
Для транскрипции в клетках, таких как бактериальные клетки, были включены последовательности промотора и терминатора, чтобы облегчить экспрессию в качестве трансгена, например, с помощью индуцируемого промотора. Длина двухцепочечной области и длина последовательности петли могут быть разными. Конструкции получали с помощью стандартных методов клонирования или заказывали у коммерческих поставщиков.
Синтез РНК
После расщепления рестрикционным ферментом для линеаризации ДНК на 3'-конце, транскрипция с использованием РНК-полимеразы привела к получению 5' и 3'-плечей транскрипта кпРНКи, отжигающегося с центральной последовательностью-мишени, причем молекула содержит центральный стебель или двухцепочечный участок с одним ником и концевыми петлями. Центральная последовательность может быть ориентирована в смысловой или антисмысловой ориентации относительно промотора (Фигура 1А, 1В соответственно).
Для in vitro синтеза, ДНК конструкции расщепляли по 3'-сайту рестрикции с помощью подходящего рестрикционного фермента, осаждали, очищали и количественно определяли. Синтез РНК выполняли с использованием РНК-полимеразы согласно инструкциям производителя. кпРНК ресуспендировали в буфере для отжига (25 мМ Трис-HCl, pH 8,0, 10 мМ MgCl2) с использованием обработанной DEPC воды для инактивации следов РНКазы. Выход и целостность РНК, полученной при использовании этого метода, определяли с помощью анализа NanoDrop и гель-электрофореза (Фигура 2), соответственно.
Синтез кпРНК обеспечивали в бактериальных клетках путем введения конструкций в штамм E. coli HT115. Трансформированные культуры клеток индуцировали IPTG (0,4 мМ) для экспрессии РНК-полимеразы T7 с обеспечением транскрипции конструкций кпРНК. Выделение РНК из бактериальных клеток и очистку проводили, как по существу описано в публикации Timmons et al. (2001).
Для транскрипции РНК с Cy3-мечением, смесь рибонуклеотидов (рНТФ) содержала по 10 мМ АТФ, ГТФ, ЦТФ, 1,625 мМ УТФ и 8,74 мМ Cy3-УТФ. Реакции транскрипции инкубировали при 37°C в течение 2,5 часов. Реакции транскрипции (160 мкл) переносили в пробирки Эппендорф, добавляли 17,7 мкл буфера turbo DNase и 1 мкл turbo DNA, и инкубировали при 37°C в течение 10 минут для расщепления ДНК. Затем добавляли 17,7 мкл инактивирующего раствора Turbo DNAse, перемешивали и инкубировали при комнатной температуре в течение 5 минут. Смесь центрифугировали в течение 2 минут и переносили супернатант в новую пробирку Эппендорф без РНКаз. Образцы по 1,5 мкл каждой реакции транскрипции подвергали гель-электрофорезу для проверки качества продукта РНК. Обычно наблюдали одну полосу РНК размером 500-1000 пн в зависимости от конструкции. РНК осаждали путем добавления в каждую пробирку: 88,5 мкл 7,5 M ацетата аммония и 665 мкл холодного 100% этанола. Пробирки охлаждали до -20°C в течение нескольких часов или в течение ночи, затем центрифугировали при 4°C в течение 30 мин. Супернатант осторожно удаляли и осадок РНК промывали 1 мл 70% этанола (приготовленнного с безнуклеазной водой) при -20°C и центрифугировали. Осадок сушили и ресуспендировали очищенную РНК в 50 мкл 1× буфера для отжига РНКи. Концентрацию РНК измеряли с помощью метода NanoDrop и хранили при -80°C до использования.
Пример 2. Конструкция кпРНК
Как схематично показано на Фигуре 1A, типичная молекула кпРНК включает смысловую последовательность, которая может считаться двумя смежными смысловыми последовательностями, ковалентно связанными и обладающими идентичностью с РНК-мишенью, антисмысловую последовательность, которая комплементарна смысловой последовательности и которая разделена на две области и две петли, отделяющие смысловую от антисмысловой последовательности. Конструкция ДНК, кодирующая эту форму кпРНК таким образом, включает, в порядке от 5' к 3', промотор для транскрипции кодирующей области кпРНК, первую антисмысловую область, обладающую комплементарностью с областью в направлении 5'-конца РНК-мишени, первую последовательность петли, смысловую последовательность, вторую последовательность петли, затем вторую антисмысловую область, обладающую комплементарностью с областью в направлении 3'-конца РНК-мишени, и наконец средство терминации транскрипции. В этой конфигурации две антисмысловых последовательности фланкировали смысловую последовательность и последовательности петли. В ходе транскрипции две области антисмысловой последовательности отжигаются со смысловой последовательностью, образуя стебель дцРНК с двумя фланкирующими петлями.
В другой, но родственной, форме кпРНК смысловая последовательность разделена на две области, тогда как две антисмысловых области остаются в виде единой последовательности (Фигура 1B). Конструкция ДНК, кодирующая эту вторую форму кпРНК, таким образом, включает, в порядке от 5' к 3', промотор для транскрипции кодирующей области кпРНК, первую смысловую область, обладающую идентичностью с областью в направлении 3'-конца РНК-мишени, первую последовательность петли, антисмысловую последовательность, вторую последовательность петли, затем вторую смысловую область, обладающую идентичностью в отношении 5'-конца РНК-мишени, и, наконец, средство терминации транскрипции. В этой конфигурации две смысловых последовательности фланкированы антисмысловой последовательностью и последовательностями петли.
Без ограничения теорией, из-за наличия замкнутых петель на каждом конце, такие структуры кпРНК будут более устойчивы к экзонуклеазам, чем дцРНК с открытыми концами, образованная между одноцепочечной смысловой и антисмысловой РНК и не имеющая петель, а также по сравнению со шпилечной РНК, имеющей только одну петлю. Кроме того, авторы изобретения предположили, что петля на обоих концах стебля дцРНК позволит Dicer эффективно получать доступ к обоим концам, улучшая, таким образом, процессинг дцРНК с образованием кРНК и повышением эффективности сайленсинга.
В качестве первого примера была создана генетическая конструкция для транскрипции in vitro с использованием РНК-полимеразы T7 или SP6 для образования кпРНК, направленных на гены, кодирующие GFP или GUS. Конструкция ledGFP содержала следующие области в порядке: первая половина антисмысловой последовательности соответствовала нуклеотидам 358-131 кодирующей последовательности GFP (CDS) (SEQ ID NO: 7), первая антисмысловая петля соответствовала нуклеотидам 130-1 CDS GFP, смысловая последовательность соответствовала нуклеотидам 131-591 CDS GFP, вторая антисмысловая петля соответствовала нуклеотидам 731-592 CDS GFP, и вторая половина антисмысловой последовательности соответствовала нуклеотидам 591-359 CDS GFP.
Конструкция ledGUS включила следующие области в порядке: первая половина антисмысловой последовательности соответствовала нуклеотидам 609-357 CDS GUS (SEQ ID NO:8); первая антисмысловая петля соответствовала нуклеотидам 356-197 CDS GUS, смысловая последовательность соответствовала нуклеотидам 357-860 CDS GUS, вторая антисмысловая петля соответствовала нуклеотидам 1029-861 CDS GUS; и вторая половина антисмысловой последовательности соответствовала нуклеотидам 861-610 CDS GUS.
Для получения отдельной цепи смысловой/антисмысловой дцРНК GUS (обычной дцРНК), такую же последовательность-мишень, соответствующую нуклеотидам 357-860 CDS GUS, лигировали между T7 и SP6 промоторами в векторе pGEM-T Easy. Смысловые и антисмысловые цепи транскрибировали отдельно с помощью T7 или SP6 полимераз, соответственно, и отжигали в буфера для отжига после смешивания транскриптов и нагревания смеси для денатурации цепей РНК.
Пример 3. Стабильность кпРНК
Способность кпРНК формировать структуры дцРНК сравнивали с дцРНК, имеющей открытые концы (т.е. не имеющей петель, сформированных при отжиге отдельной одноцепочечной смысловой и антисмысловой РНК), и длинной шРНК. кпРНК, длинную шРНК и смесь смысловой и антисмысловой РНК денатурировали при кипячении и позволяли отжигаться в буфера для отжига (250 мМ Трис-HCl, pH 8,0 и 100 мМ MgCl2), а затем подвергали электрофорезу в 1,0% агарозном геле в неденатурирующих условиях.
Как показано на Фигуре 2, и GUS кпРНК, и GFP кпРНК давали доминирующую полосу РНК с подвижностью, ожидаемой для двухцепочечной молекулы, что указывало на образование ожидаемой структуры кпРНК. Это отличалось от смеси смысловой и антисмысловой РНК, показавшей лишь слабую полосу для дцРНК, что указывало на то, что большая часть смысловых и антисмысловых РНК не отожигались друг с другом, образуя дцРНК. Образцы шпилечной РНК давали две заметных полосы, что указывало на то, что только часть транскрипта формировала ожидаемую структуру шпилечной РНК. Таким образом, кпРНК оказалась наиболее эффективной при формировании ожидаемой структуры дцРНК.
Способность кпРНК оставаться и распространяться на поверхности листа также сравнивали с дцРНК. GUS кпРНК (ledGUS), при нанесении на нижнюю часть поверхности листа табака, можно было легко обнаружить в необработнной верхней части листа через 24 часа (Фигура 3). Однако отдельная цепь GUS дцРНК (dsGUS) не была обнаружена в необработнной верхней части листа (Фигура 3). Этот результат указывает, что кпРНК более устойчива к деградации, чем дцРНК, и поэтому способна распространяться внутрь тканей листьев растения.
Пример 4. Тестирование кпРНК путем наружной доставки
Способность кпРНК вызывать РНКи после наружной доставки тестировали в растениях Nicotiana benthamiana и Nicotiana tabacum, экспрессирующие репортерный ген GFP или GUS, соответственно. Последовательности GFP и GUS последовательностей-мишеней и кодирующие кпРНК конструкции показаны в SEQ ID NO: 7, 8, 4 и 5, соответственно. Рибонуклеотидная последовательность кодируемых молекул РНК представлена в SEQ ID NO:1 (GFP кпРНК) и 2 (GUS кпРНК).
Для облегчения воспроизводимого и равномерного нанесения кпРНК на поверхности листьев, кпРНК в концентрации 75-100 мкг/мл в 25 мМ Трис-HCl, pH 8,0, 10 мМ MgCl2 и Silwet 77 (0,05%) наносили на адаксиальную поверхность листьев с помощью мягкой кисти. Через 6 часов и 3 дня после нанесения кпРНК образцы листьев собирали для анализа направленного сайленсинга гена-мишени.
Нанесение кпРНК против GFP в листьях N. benthamiana и против GUS в листьях N. tabacum привело к четкому снижению на 20-40% и 40-50% соответствующей активности гена-мишени на уровне активности мРНК (GFP) или белка (GUS) через 6 часов после обработки. Однако в этом эксперименте снижение не сохранялось через 3 дня после обработки. Авторы изобретения предположили, что такое наблюдение через 3 дня было, вероятно, связано с некоторыми неспецифическими реакциями трансгенов на обработку дцРНК или рассеивания количества кпРНК. Однако в отдельном эксперименте сайленсинг GUS был обнаружен как на обработанных, так и на дистальных необработанных участках листьев через 24 часа после обработки кпРНК (Фигура 4).
Пример 5. кпРНК-индуцированный сайленсинг эндогенного гена-мишени
В следующем примере кпРНК была разработана для направленного воздействия на мРНК, кодируемую эндогенным геном, а именно геном FAD2.1 N. benthamiana. Последовательности мРНК-мишени FAD2.1 и кодирующей конструкции ledFAD2.1 показаны в SEQ ID NO: 9 и 6 соответственно. Рибонуклеотидная последовательность молекулы кодируемой РНК представлена в SEQ ID NO: 3 (кпРНК FAD2.1 N. benthamiana).
Конструкция кпРНК FAD2.1 состояла из следующего: первая половина антисмысловой последовательности, соответствующей нуклеотидам 678-379 CDS FAD2.1 (Niben101Scf09417g01008.1); первая антисмысловая петля, соответствующая нт 378-242 CDS FAD2.1; смысловая последовательность, соответствующая нт 379-979; вторая антисмысловая петля, соответствующая нт 1115-980; и вторая половина антисмысловой последовательности, соответствующая нт 979-679 CDS FAD2.1.
РНК ledGUS из предыдущего примера использовали параллельно в качестве отрицательного контроля. В первом эксперименте сайленсинг гена-мишени анализировали по уровню мРНК FAD2.1 и по накоплению жирной кислоты C18:1 (Фигура 5). Также анализировали уровень активности родственного гена, FAD2.2. Для каждого образца приблизительно 3 мкг суммарную РНК обрабатывали ДНКазой и обратно транскрибировали при 50°C в течение 50 минут с использованием олиго-дТ праймеров. Реакции останавливали при 85°C в течение 5 минут и разбавляли водой до 120 мкл. С помощью ПЦР-амплификатора Rotor Gene по 5 мкл каждого образца в трех экземплярах исследовали на их относительную экспрессию мРНК FAD 2.1 и FAD 2.2 с использованием геноспецифических праймеров с контролем по гену домашнего хозяйства актину. В последующем эксперименте для подтверждения сайленсинга гена FAD2.1 путем наружного применения РНК ledFAD2.1 также использовали нозерн-блот гибридизацию (Фигура 6).
мРНК FAD2.1 была значимо понижена до уровня, который почти не обнаруживался в тканях листьев, обработанных кпРНК, в моменты времени 2, 4 и 10 часов (Фигура 5). В этом эксперименте было неясно, почему уровень мРНК FAD2.1 не уменьшился так сильно через 6 часа. В повторном эксперименте, показанном на Фигуре 6, сильная даунрегуляция FAD2.1 наблюдалась как через 6, так и через 24 часа, особенно в точке времени 24 часа. Родственный ген FAD2.2, с гомологией последовательности с FAD2.1, также демонстрировал даунрегуляцию в точках времени 2 и 4 часа при воздействии кпРНК (Фигура 5).
Поскольку FAD2.1 и FAD2.2 кодируют Δ12-десатуразы жирных кислот, которые десатурируют олеиновую кислоту до линолевой кислоты, уровни этих жирных кислот исследовали в тканях листьев, обработанных кпРНК. Наблюдали четкое увеличение накопления олеиновой кислоты (18:1) в тканях листьев, обработанных кпРНК, в точки времени 2, 4 и 6 часов, что указывало на уменьшение количества фермента FAD2 (Фигура 5). Таким образом, как кОТ-ПЦР, так и анализ состава жирных кислот показали, что кпРНК индуцирует сайленсинг гена FAD2.1.
Пример 6. Конструкция и тестирование шпилечных РНК, включающих пары оснований G:U или неспаренные нуклеотиды
Модифицированные шпилечные РНК, направленно воздействующие на РНК GUS
Репортерные гены, такие как ген, кодирующий фермент β-глюкуронидазу (GUS), обеспечивают простую и удобную систему анализа, которую можно применять для измерения эффективности сайленсинга генов в эукариотической клетке, в том числе в клетках растений (Jefferson et al., 1987). Поэтому авторы изобретения разработали, получили и протестировали некоторые модифицированные шпилечные РНК на их способность снижать экспрессию гена GUS в качестве гена-мишени, используя подход доставки генов для введения шпилечных РНК в клетки, и сравнили модифицированные шпильки с обычной шпилечной РНК. Обычная шпилечная РНК, используемая в качестве контроля в эксперименте, имела двухцепочечную область длиной 200 последовательных пар оснований, в которой все пары оснований были каноническими парами оснований, то есть парами оснований G:C и A:U без пар оснований G:U, и без каких-либо неспаренных нуклеотидов (некомплементарных оснований) в двухцепочечной области, направленную на такую же 200 нт область молекулы мРНК GUS, что и модифицированные шпилечные РНК. Смысловые и антисмысловые последовательности, которые образовывали двухцепочечную область, были ковалентно связаны спейсерной последовательностью, включающей интрон PDK (Helliwell et al., 2005; Smith et al., 2000), с получением РНК-петли длиной 39 или 45 нуклеотидов (в зависимости от используемой стратегии клонирования) после сплайсинга интрона из первичного транскрипта. Фрагмент ДНК, используемый для антисмысловой последовательности, был фланкирован сайтами рестрикции XhoI-BamHI на 5'-конце и сайтами рестрикции HindIII-KpnI на 3'-конце для легкого клонирования в кассету экспрессии, и каждая смысловая последовательность была фланкирована сайтами рестрикции XhoI и KpnI. Область дцРНК размером 200 пн каждой шпилечной РНК, как для контрольной шпильки, так и для модифицированных шпилек, включала антисмысловую последовательность из 200 нуклеотидов, которая была полностью комплементарна последовательности GUS дикого типа в кодирующей белок области. Эта антисмысловая последовательность, соответствующая нуклеотидам 13-212 SEQ ID NO:10, являлась комплементом нуклеотидов 804-1003 открытой рамки считывания GUS (ORF) (последовательность кДНК предоставлена в SEQ ID NO:8). Таким образом, мРНК-мишень GUS имела длину больше 1900 нт. Длина 200 нуклеотидов для смысловой и антисмысловой последовательностей была выбрана как достаточно малая, чтобы она была достаточно удобной для синтеза фрагментов ДНК с использованием синтетических олигонуклеотидов, но также достаточно длинная, чтобы давать множество молекул рРНК при процессинге Dicer. Являясь частью ORF, последовательность, по-видимому, не содержала криптические сайты сплайсинга или сайты терминации транскрипции.
Получение генетических конструкций
Последовательность 200 пн ORF GUS амплифицировали с помощью ПЦР при использовании пары олигонуклеотидных праймеров GUS-WT-F (SEQ ID NO:52) и GUS-WT-R (SEQ ID NO:53), содержащих сайты XhoI и BamHI или сайты HindIII и KpnI, соответственно, для введения сайтов этих рестриктаз 5' и 3' от последовательности GUS. Амплифицированный фрагмент встраивали в вектор pGEM-T Easy и правильную нуклеотидную последовательность подтверждали секвенированием. Фрагмент GUS вырезали путем расщепления BamHI и HindIII и встраивали по сайтам BamHI/HindIII pKannibal (Helliwell and Waterhouse, 2005), что давало вставку последовательности GUS в антисмысловой ориентации относительно функционально связанного e35S промотора CaMV (Grave, 1992) и терминатора полиаденилирования/транскрипции гена ocs (Ocs-T). Полученный вектор был обозначен pMBW606 и содержал, в порядке от 5' к 3', 35S::PDK Интрон::антисмысловой GUS::кассету экспрессии Ocs-T. Этот вектор был промежуточным вектором, который использовали в качестве базового вектора для сборки четырех конструкций шРНК.
Конструкция hpGUS[wt], содержащая только канонические пары оснований
Для получения вектора, обозначенного hpGUS[wt], кодирующего молекулу шпилечной РНК, используемую в качестве контроля в эксперименте, содержащую только канонические пары оснований, ПЦР-фрагмент GUS длиной 200 пн вырезали из плазмиды pGEM-T Easy с помощью XhoI и KpnI и встраивали по сайтам XhoI/KpnI между 35S промотором и интроном PDK в pMBW606. В результате получали вектор, обозначенный pMBW607, содержащий кассету экспрессии 35S::смысловой GUS[wt]::интрон PDK::антисмысловой GUS::OCS-T. Эту кассету вырезали путем расщепления NotI и встраивали в сайт NotI pART27 (Gleave, 1992) с получением в результате вектора, обозначенного hpGUS[wt], кодирующего канонически спаренную шпилечную РНК, направленно воздействующую на мРНК GUS.
При отжиге на себя в результате гибридизации 200 нт смысловой и антисмысловой последовательностей эта шпилька содержала двухцепочечную область из 200 последовательных пар оснований, соответствующих последовательностям GUS. Каждая из смысловой и антисмысловой последовательностей в кассете экспрессии была фланкирована сайтами рестрикции BamHI и HindIII, присутствующими на 5' и 3'-концах, соответственно, по отношению к смысловой последовательности GUS. При транскрипции нуклеотиды, соответствующие этим сайтам, также могли гибридизоваться, удлиняя двухцепочечную область на 6 пн на каждом конце. После транскрипции кассеты экспрессии и сплайсинга интрона PDK из первичного транскрипта предположили, что структура шпилечной РНК перед каким-либо процессингом Dicer или другими РНКазами будет иметь петлевую структуру из 39 нуклеотидов. Нуклеотидная последовательность структуры шпилечной РНК, включая ее петлю, представлена в SEQ ID NO:15, при этом ее свободная энергия свертывания по расчетам должна была составлять -471,73 ккал/моль. Следовательно, это была энергетически стабильная шпилечная структура. Свободную энергию вычисляли при использовании "RNAfold" (http://rna.tbi.univie.ac.at/cgi-bin/RNAWebSuite/RNAfold.cgi) на основе нуклеотидных последовательностей после сплайсинга последовательности интрона PDK.
При транскрипции с кассеты экспрессии, содержащей 35S промотор и терминатор OCS-T, полученные шпилечные РНК встраивали в более крупную молекулу РНК с добавлением 8 нуклеотидов на 5'-конец и добавлением приблизительно 178 нуклеотидов на 3'-конец, без учета добавления какого-либо поли(A)-хвоста на 3'-конец. Поскольку один и тот же дизайн промотора-терминатора использовали для модифицированных шпилечных РНК, эти молекулы также имели такие удлинения на 5' и 3'-концах. Таким образом, длина молекулы шпилечной РНК после сплайсинга интрона PDH составляла приблизительно 630 нуклеотидов.
Конструкции hpGUS[G:U], включающие пары оснований G:U
Сборку фрагмента ДНК, содержащего такую же смысловую последовательность из 200 нуклеотидов, но в которой все 52 цитидин-нуклеотида (С) соответствующей области GUS дикого типа были заменены тимидиновыми нуклеотидами (Т), проводили путем отжига перекрывающихся олигонуклеотидов GUS-GU-F (SEQ ID NO:54) и GUS-GU-R (SEQ ID NO:55) и ПЦР достройки 3'-концов с использованием высокоточной Taq-полимеразы LongAmp (New England Biolabs, номер по каталогу M0323). Амплифицированный фрагмент ДНК встраивали в вектор pGEM-T Easy и правильную нуклеотидную последовательность (SEQ ID NO:11) подтверждали секвенированием. Затем фрагмент ДНК, содержащий модифицированную последовательность, вырезали путем расщепления XhoI и KpnI и встраивали по сайтам XhoI/KpnI базового вектора pMBW606. В результате была получена конструкция, обозначенная pMBW608, содержащая кассету экспрессии 35S::смысловой GUS[G:U]::интрон PDK::антисмысловой GUS::OCS-T. Эту кассету экспрессии вырезали путем расщепления NotI и встраивали в сайт NotI pART27 с получением в результате вектора, обозначенного hpGUS[G:U], кодирующего G:U спаренную молекулу шпилечной РНК.
Эта кассета кодировала шпилечную РНК, направленную на мРНК GUS, и которая при отжиге на себя путем гибридизации 200 нт смысловой и антисмысловой последовательностей содержала 52 пары оснований G:U (вместо пар оснований G:C в hpGUS[wt]) и 148 канонических пар оснований, то есть 26% нуклеотидов двухцепочечной области были включены в пары оснований G:U. 148 канонических пар оснований в hpGUS[G:U] были такими же, как в контрольной шпилечной РНК, в соответствующих положениях, включая 49 пар оснований U:A, 45 пар оснований A:U и 54 пары оснований G:C. Наиболее длинные непрерывные отрезки канонических пар оснований в двухцепочечной области содержали 9 пар оснований. Таким образом, антисмысловая нуклеотидная последовательность hpGUS[G:U] имела идентичную длину (200 нт) и последовательность с антисмысловой последовательностью контрольной шпилечной РНК hpGUS[wt]. После транскрипции кассеты экспрессии и сплайсинга интрона PDK из первичного транскрипта ожидали, что структура шпилечной РНК перед процессингом Dicer или другими РНКазами будет содержать петлевую структуру из 45 нуклеотидов. Нуклеотидная последовательность шпилечной структуры, включая ее петлю, представлена в SEQ ID NO:16, и ее свободная энергия свертывания по расчетам должна была составлять -331,73 ккал/моль. Что касается hpGUS[wt], то это была энергетически стабильная шпилечная структура, несмотря на присутствие 52 пар оснований G:U, которые по отдельности намного слабее, чем пары оснований G:C в hpGUS[wt].
Выравнивание модифицированной смысловой последовательности GUS (нуклеотиды 9-208 SEQ ID NO:11) с соответствующей областью гена-мишени GUS (SEQ ID NO:14) показано на Фигуре 7.
Конструкции hpGUS[1:4], включающие некоплементарные нуклеотиды в каждом четвертом положении
Был разработан и собран фрагмент ДНК, включающий такую же 200 пн смысловую последовательность, но в которой заменяли каждый четвертый нуклеотид соответствующей последовательности GUS дикого типа. Каждый 4-й нуклеотид в каждом блоке из 4 нуклеотидов (нуклеотиды в положениях 4, 8, 12, 16, 20 и т.д.) заменяли путем замены C на G, G на C, A на T и T на A, другие нуклеотиды оставляли без изменений. Все эти замены были трансверсными заменами, которые, как ожидали, будут оказывать более сильный дестабилизирующий эффект на полученную в результате структуру шпилечной РНК, чем транзиционные замены. Сборку фрагмента ДНК проводили путем отжига перекрывающихся олигонуклеотидов GUS-4M-F (SEQ ID NO:56) и GUS-4M-R (SEQ ID NO:57) и ПЦР достройки 3'-концов с помощью Taq-полимеразы LongAmp. Амплифицированный фрагмент ДНК встраивали в вектор pGEM-T Easy и правильную нуклеотидную последовательность (SEQ ID NO:12) подтверждали секвенированием. Фрагмент ДНК, включающий модифицированную последовательность, затем вырезали путем расщепления XhoI и KpnI и встраивали по сайтам XhoI/KpnI базового вектора pMBW606. В результате получали конструкцию, обозначенную pMBW609, содержащую кассету экспрессии 35S::смысловой GUS[1:4]::интрон PDK::антисмысловой GUS::OCS-T. Эту кассету экспрессии вырезали при расщеплении NotI и встраивали в сайт NotI pART27 с получением вектора, обозначенного hpGUS[1:4], кодирующего молекулу шпилечной РНК, содержащую 1:4 некомплементарное основание.
Эта кассета кодировала шпилечную РНК, направленную на мРНК GUS, и которая при отжиге на себя путем гибридизации смысловой и антисмысловой последовательностей содержала 50 некомплементарных нуклеотидов из 200 нт антисмысловой последовательности, включая некомплементарное основание нуклеотида в положении 200. За исключением положения 200, двухцепочечная область шпилечной РНК содержала 150 канонических пар оснований и 49 некомплементарных нуклеотидных пар на протяжении длины 199 нт смысловой и антисмысловой последовательностей, т.е. ожидали, что 24,6% нуклеотидов двухцепочечной области будут некомплементарны друг другу (не будут включены в пары оснований). После транскрипции кассеты экспрессии и сплайсинга интрона PDK из первичного транскрипта ожидали, что структура шпилечной РНК перед процессингом Dicer или другими РНКазами будет содержать петлевую структуру из 45 нуклеотидов. Нуклеотидная последовательность шпилечной структуры, включая ее петлю, представлена в SEQ ID NO:17, и ее свободная энергия свертывания по расчетам должна составлять -214,05 ккал/моль. Что касается hpGUS[wt], то это была энергетически стабильная шпилечная структура, несмотря на некомплементарные нуклеотиды.
Выравнивание модифицированной смысловой последовательности GUS (нуклеотиды 9-208 SEQ ID NO:12) с соответствующей областью гена-мишени GUS (SEQ ID NO:14) показано на Фигуре 8.
Конструкции hpGUS[2:10], в которых нуклеотиды 9 и 10 из 10 нуклеотидов юыли некомплементарными
Выполняли разработку и сборку фрагмента ДНК, включающего такую же 200 пн смысловую последовательность, но в котором каждый девятый и десятый нуклеотид соответствующей последовательности GUS дикого типа был заменен. Каждый 9-й и 10-й нуклеотид в каждом блоке из 10 нуклеотидов (нуклеотиды в положениях 9, 10, 19, 20, 29, 30 и т.д.) заменяли путем замены C на G, G на C, A на T и T на A, другие нуклеотиды оставляли без изменений. Фрагмент ДНК собирали путем отжига перекрывающихся олигонуклеотидов GUS-10M-F (SEQ ID NO:58) и GUS-10M-R (SEQ ID NO:59) и ПЦР достройки 3'-концов с помощью Taq-полимеразы LongAmp. Амплифицированный фрагмент ДНК встраивали в pGEM-T Easy и правильную нуклеотидную последовательность (SEQ ID NO:13) подтверждали секвенированием. Фрагмент ДНК, включающий модифицированную последовательность, затем вырезали путм расщепления XhoI и KpnI и встраивали по сайтам XhoI/KpnI базового вектора pMBW606. В результате получали конструкцию, обозначенную pMBW610, содержащую кассеты экспрессии 35S::смысловой GUS[2:10]::интрон PDK::антисмысловой GUS::OCS-T. Эту кассету экспрессии вырезали при расщеплении NotI и встраивали в сайт NotI pART27 с получением вектора, обозначенного hpGUS[2:10], кодирующего молекулу шпилечной РНК, содержащую 2:10 некомплементарных основания.
Эта кассета кодировала шпилечную РНК, направленную на мРНК GUS, которая при отжиге на себя путем гибридизации смысловой и антисмысловой последовательностей содержала 50 некомплементарных нуклеотидов из 200 нт антисмысловой последовательности, включая некомплементарные основания нуклеотидов в положениях 199 и 200. За исключением положений 199 и 200, двухцепочечная область шпилечной РНК содержала 160 канонических пар оснований и 19 некомплементарных динуклеотидов на протяжении длины 198 нт смысловой и антисмысловой последовательностей, т.е. ожидали, что 19,2% нуклеотидов двухцепочечной области будут некомплементарны друг другу (не включены в пары оснований). 160 пар оснований в hpGUS[2:10] были такими же, как в контрольной шпилечной РНК, в соответствующих положениях, включая 41 пару оснований U:A, 34 пары оснований A:U, 42 G:C и 43 C:G пары оснований. После транскрипции кассеты экспрессии и сплайсинга интрона PDK из первичного транскрипта ожидали, что структура шпилечной РНК перед процессингом Dicer или другими РНКазами будет содержать петлевую структуру из 45 нуклеотидов. Нуклеотидная последовательность шпилечной структуры, включая ее петлю, представлена в SEQ ID NO:18, и ее свободная энергия свертывания по расчетам составляет -302,78 ккал/моль. Что касается hpGUS[wt], то это была энергетически стабильная шпилечная структура, несмотря на некомплементарные нуклеотиды, которые, как ожидалось, выступали из стебля шпилечной структуры.
Выравнивание модифицированной смысловой последовательности GUS (нуклеотиды 9-208 SEQ ID NO:13) с соответствующей областью гена-мишени GUS (SEQ ID NO:14) показано на Фигуре 9.
Четыре генетических конструкции для экспрессии контрольной и модифицированной шпилечных РНК схематично показано на Фигуре 10.
Пример 7. Тестирование модифицированных шпилечных РНК в трансгенных растениях
Растения вида Nicotiana tabacum (табак), трансформированные геном-мишенью GUS, использовали для тестирования эффективности четырех конструкций шпилечных РНК, описанных выше. В частности, растения-мишени были из двух гомозиготных, независимых трансгенных линий, PPGH11 и PPGH24, каждая из которых содержала однокопийную вставку трансгена GUS из вектора pWBPPGH, который схематически показан на Фигуре 11. Ген GUS в Т-ДНК pWBPPGH имел кодирующую область GUS (нуклеотиды 7-1812 SEQ ID NO:8), функционально связанную с промотором длиной 1,3 тпн гена белка флоэмы 2 (PP2) из Cucurbita pepo L. cv. Autumn Gold (Wang et al., 1994; Wang, 1994). Конструкцию pWBPPGH получали путем вырезания промотора PP2 плюс 5'-UTR и 54 нуклеотидов кодирующей области белка PP2, кодирующих первые 18 аминокислот PP2, из лямбда-геномного клона CPP1.3 (Wang, 1994) и слияния этого фрагмента с кодирующей последовательностью GUS, начиная с нуклеотидов, кодирующих 3-ю аминокислоту GUS, с получением слитого на N-конце полипептида, обладающего активностью GUS. Кассету pPP2::GUS:Nos-T встраивали в pWBVec2a (Wang et al., 1998) с получением pWBPPGH, который использовали для трансформации растений Nicotiana tabacum cv. Wisconsin 38 при использовании опосредованной Agrobacterium tumefaciens трансформации листовых дисков (Ellis et al., 1987), отбора по устойчивости к гигромицину. Активность GUS у гомозиготных растений потомства двух трансгенных линий PPGH11 и PPGH24 была сходной. Экспрессия GUS в обоих трансгенных растениях не ограничивалась флоэмой, но присутствовала в большей части тканей растений. Следовательно, экспрессия GUS с промотора PP2 в этих растениях, по-видимому, является конститутивной. Имелись две причины выбора растений PP2-GUS в качестве тестируемых растений: i) они дают относительно высокие уровни экспрессии GUS, примерно такие же, как у растений 35S-GUS; ii) промотор PP2 является эндогенным промотором гена PP2, полученным из Cucurbita pepo, с последовательностью, отличной от промотора 35S, используемого для направления экспрессии шРНК трансгенов, которые, таким образом, не будут подвергаться транскрипционной косупрессии входящим 35S промотором.
Все четыре конструкции шпилечных РНК (Пример 6) использовали для трансформации растений PPGH11 и PPGH24 при использовании Agrobacterium-опосредованного метода с листовыми дисками (Ellis et al., 1987), используя 50 мг/л канамицина в качестве селективного агента. Было обнаружено, что эта система отбора с канамицином, агентом, отличающимся от используемого ранее гигромицина, использованного для введения Т-ДНК pWBPPGH, дает только трансформированные растения без регенерации нетрансформированных растений. Регенерированные трансгенные растения, содержащие T-ДНК из конструкций hpGUS, переносили в почву для выращивания в теплице и выдерживали в течение примерно 4 недель перед анализом активности GUS. При анализе трансгенные растения были здоровыми, активно росли и по внешнему виду были идентичны нетрансформированным контрольным растениям и исходным растениям PPGH11 и PPGH24. Всего было получено 59 трансгенных растений, которые трансформировали Т-ДНК, кодирующей hpGUS[wt], были получены 74 растения, которые были трансформированы Т-ДНК, кодирующей hpGUS[G:U], 33 растения, которые были трансформированы T-ДНК, кодирующей hpGUS[1:4], и 41 растение, которые были трансформированы T-ДНК, кодирующей hpGUS[2:10].
Уровни экспрессии GUS измеряли с помощью флуориметрического анализа с 4-метилумбеллиферил-β-D-глюкуронида (MUG) (Jefferson et al., 1987), согласно модифицированному кинетическому методу, описанному в Chen et al. (2005). Растения анализировали, собирая образцы листьев диаметром примерно 1 см из трех разных листьев на каждом растении, выбирая листья, которые были хорошо развернутыми, здоровыми и зелеными. Были предприняты меры для того, чтобы исследуемые растения находились на одной стадии роста и развития, что и контрольные растения. В каждом анализе использовали по 5 мкг белка, выделенного из каждого образца листа, и измеряли скорость расщепления MUG, как описано в Chen et al. (2005).
Репрезентативные данные показаны на Фигуре 12, на которой показана активность GUS (единицы MUG в анализе) для каждого независимого трансгенного растения. Поскольку данные для конструкции hpGUS[wt] показали, что некоторые растения демонстрировали сильный сайленсинг с уменьшением активности по меньшей мере на 90%, а другие - более слабый сайленсинг, 10% активность GUS по сравнению с контрольными растениями в этом контексте была выбрана в качестве уровня активности для разделения растений на две категории и сравнения разных конструкций.
Генетическая конструкция, кодирующая hpGUS[wt] с канонически спаренными основаниями, индуцировала сильный сайленсинг GUS, при использовании уровня 10% активности в качестве критерия сильного сайленсинга, в 32 из 59 протестированных трансгенных растений (54,2%). Остальные 27 растений показали пониженную активность GUS, но сохранили больше 10% активности фермента по сравнению с контрольными растениями, и поэтому считалось, что они демонстрируют слабый сайленсинг в этом контексте. Трансгенные растения с этой конструкцией демонстрировали широкий диапазон уровня сайленсинга гена GUS (Фигура 12), от активности меньше 1% до приблизительно 80% остаточной активности, что было типично для обычных шпилечных конструкций (Smith et al., 2000).
В отличие от этого, конструкция hpGUS[G:U] индуцировала однородный и единообразный сайленсинг в разных независимых трансгенных линиях, при этом 71 из 74 растений (95,9%), которые были протестированы, демонстрировали сильный сайленсинг GUS. И опять же, в отличие от этого все 33 протестированных растения hpGUS[1:4] показали сниженные уровни активности GUS, причем только 8 (24%) давали <10% активности GUS по сравнению с контрольными растениями, а остальные 25 классифицировали как имеющие более слабый сайленсинг. Эти результаты показали, что эта конструкция индуцировала более слабые, но более единообразные уровни даунрегуляции GUS в трансгенных линиях. Конструкция hpGUS[2:10] действовала более подобно конструкции hpGUS[wt], индуцируя хорошие уровни сайленсинга в некоторых линиях (28 из 41, или 68,3%), и вызывала слабый или вообще не вызывала сайленсинга GUS в остальных 13 растениях.
Когда для сравнения использовали только сайленсированные линии (остаточная активность <10%) и вычисляли среднюю активность GUS, растения hpGUS[wt] продемонстрировали наиболее высокую среднюю степень сайленсинга, после чего следовали по порядку растения hpGUS[G:U] и растения hpGUS [2:10] (Фигура 13). Растения hpGUS[1:4] показали наименьшее среднее снижение активности GUS. Степень сайленсинга GUS показала хорошую корреляцию с термодинамической стабильностью предсказанных структур шРНК, полученных из четырех различных конструкций шРНК (Пример 6).
Для проверки, сохранятся ли различия у растений потомства, репрезентативные трансгенные растения, содержащие как ген-мишень GUS, которые были гомозиготными, так и трансген hpGUS (гемизиготные), самоопыляли. Были отобраны устойчивые к канамицину растения потомства из линий hpGUS, поэтому отбрасывали любые нулевые сегреганты, в которых отсутствовали трансгены hpGUS. Это гарантировало, что трансгены hpGUS присутствовали во всем потомстве, в гомозиготном или гетерозиготном состоянии. Растения потомства исследовали на активность GUS, репрезентативные данные представлены на Фигуре 14. Потомство, содержащее трансгены hpGUS[wt], четко разделялось на две категории, а именно растения, которые имели сильный сайленсинг GUS, и другие, которые демонстрировали слабый сайленсинг или его отсутствие. Эти классы хорошо коррелировали с фенотипом предыдущего поколения, показав, что степень сайленсинга гена-мишени была наследуемой. Все растения в протестированных линиях hpGUS[G:U] постоянно демонстрировали сильный сайленсинг, тогда как растения в линиях hpGUS[1:4] постоянно демонстрировали более слабый сайленсинг. Авторы изобретения пришли к выводу, что фенотипы, наблюдаемые в родительском поколении, обычно сохранялись в растениях потомства.
Эксперименты по Саузерн-блот гибридизации с трансформированными растениями
Однородность сильного сайленсинга генов, наблюдаемая в большом количестве независимых трансгенных растений, полученных с конструкцией hpGUS[G:U], была замечательной, а также удивительной и неожиданной. Авторы изобретения стремились установить, присутствовало ли какое-нибудь другое объяснение однородности сайленсинга кроме эффекта, вызванного РНК hpGUS[G:U]. Чтобы проверить, было ли множество трансгенных растений получено в результате независимых событий трансформации, как и предполагали, проводили эксперименты по Саузерн-блот гибридизации с ДНК, выделенной из 18 репрезентативных трансгенных растений, содержащих конструкцию hpGUS[G:U]. ДНК выделяли из тканей листьев при использовании метода горячего фенола, описанного Wang et al. (2008). Для Саузерн-блот гибридизации приблизительно 10 мкг ДНК из каждого образца растения расщепляли ферментом HindIII, разделяли с помощью гель-электрофореза в 1% агарозном геле в буфере TBE и переносили на мембрану Hybond-N+ с использованием капиллярного метода (Sambrook et al., 1989). Мембрану гибридизовали в течение ночи при 42°C с 32P-меченным фрагментом ДНК из области терминатора OCS-T. Этот зонд выбрали, поскольку он гибридизовался с трансгеном hpGUS[G:U], но не гибридизовался с геном-мишенью GUS, который не содержал последовательность терминатора OCS-T. Мембрану промывали в условиях высокой жесткости и связавшийся зонд визуализировали с помощью PhosphoImager.
Авторадиограф гибридизованного блота показан на Фигуре 15. Каждая полоса показала от одной до пяти или шести гибридизованных полос. Не было двух дорожек, демонстрирующих одинаковую картину, то есть авторадиография показала, что каждое из 16 репрезентативных растений hpGUS[G:U] имело разные паттерны фрагментов HindIII, которые гибридизовались и, следовательно, были получены из разных вставок трансгена. Авторы изобретения пришли к выводу, что единообразный сайленсинг GUS, наблюдаемый для линий hpGUS[G:U], не был обусловлен сходными паттернами вставки трансгена в растениях, и что однородность сайленсинга была вызвана структурой РНК hpGUS[G:U]. Авторы изобретения также пришли к выводу, что для получения сильного сайленсинга генов не требовалось множество копий трансгена hpGUS[G:U]; одной копии трансгена было достаточно.
Эксперименты по Нозерн-блот гибридизации с трансформированными растениями
Для определения, была ли РНК hpGUS[G:U] процессирована так же, как и контрольная шпилечная РНК в трансгенных растениях, проводили эксперименты по Нозерн-блот гибридизации с РНК, выделенной из листьев трансгенных растений. Эксперименты по Нозерн-блоттингу проводили для обнаружения более коротких РНК (кРНК длиной приблизительно 21-24 нуклеотида), которые образовывались в результате процессинга Dicer шпилечной РНК. Эксперимент проводили на малой РНК, выделенной из трансгенных растений hpGUS[wt] и hpGUS[G:U], которые также содержали ген-мишень GUS, который экспрессировался в виде (смысловой) мРНК. Для анализа кРНК были выбраны по девять растений с каждой конструкцией. Для формирования hpGUS[wt] трансгенной популяции включали растения, демонстрирующие слабый сайленсинг GUS, а также некоторые растения, демонстрирующие сильный сайленсинг GUS. Образцы малой РНК выделяли при использовании метода с горячим фенолом (Wang et al., 2008), Нозерн-блот гибридизацию проводили согласно Wang et al. (2008), а гель-электрофорез образцов РНК проводили в денатурирующих условиях. В качестве зондов использовали 32Р-меченые РНК, соответствующие либо смысловой последовательности, либо антисмысловой последовательности, соответствующей нуклеотидам 804-1003 SEQ ID NO:8.
Авторадиограф Нозерн-блоттинга, гибридизованного либо с антисмысловым зондом (верхняя панель) для обнаружения смысловых молекул кРНК, полученных из шпилечных РНК, либо гибридизованного со смысловым зондом для обнаружения антисмысловых кРНК (нижняя панель), показан на Фигуре 16. Внизу на Фигуре показана качественная оценка уровня экспрессии GUS по сравнению с контрольными растениями, в которых отсутствовали конструкции hpGUS. Наблюдали гибридизацию с малыми РНК длиной приблизительно 20-25 нуклеотидов, основанную на подвижности кРНК по сравнению с РНК известной длины в других экспериментах. Полосы hpGUS[wt] показали диапазон изменения количества накопления кРНК. Это наблюдали как для смысловой, так и для антисмысловой рРНК, хотя полосы антисмысловой рРНК были не такими четкими, как смысловые полосы. Поскольку растения hpGUS[wt] содержали как трансген hpGUS, экспрессирующий смысловые и антисмысловые последовательности, соответствующие 200 нт области-мишени, так и ген-мишень GUS, экспрессирующий полноразмерный смысловой ген, смысловые кРНК могли быть получены из любой шпилечной РНК или мРНК-мишени. По-видимому, между уровнем рРНК и степенью сайленсинга GUS в растениях hpGUS[wt] присутствует отрицательная корреляция. Например, два растения, представленные на дорожках 4 и 5, накапливали относительно больше кРНК, но демонстрировали лишь умеренную степень даунрегуляции GUS. Напротив, два растения, представленные на дорожках 7 и 8, показали сильный сайленсинг GUS, но накапливали относительно низкие уровни кРНК.
В отличие от растений hpGUS[wt] и в соответствии с относительно единообразной степенью сайленсинга под действием конструкции hpGUS[G:U], растения hpGUS[G:U] накапливали единообразное количество антисмысловых кРНК по всем полосам. Кроме того, степень сайленсинга GUS, по-видимому, показывает хорошую корреляцию с количеством антисмысловой кРНК. В этих растениях практически не было обнаружено смысловых кРНК. Это ожидали, так как РНК-зонд, используемый в Нозерн-блоттинге, транскрибировался с последовательности GUS дикого типа и, следовательно, обладал более низким уровнем комплементарности по отношению к смысловым кРНК из hpGUS[G:U], где все C нуклеотиды были заменены U нуклеотидами, допуская только гибридизацию с более низкой жескостью. Тем не менее, данный эксперимент не исключил возможности того, что РНК hpGUS[G:U] процессировалась с получением меньшего количества смысловых кРНК, или что они быстрее разрушались.
Эксперимент по Нозерн-блот гибридизации повторяли, на этот раз с использованием только смыслового зонда для обнаружения антисмысловых рРНК; авторадиограф показан на Фигуре 17. Опять же, продукция антисмысловых рРНК с конструкции hpGUS[wt] отрицательно коррелирует с активностью GUS (верхняя панель на Фигуре 17). Растения, которые подверглись сильному сайленсингу, давали высокие уровни антисмысловых кРНК (дорожки 1, 3, 5, 8 и 10), тогда как растения, которые демонстрировали лишь слабый сайленсинг или отсутствие сайленсинга, не давали сигнал гибридизации в этом эксперименте (дорожки 2, 4, 6, 7 и 9). В явной противоположности от этого, растения, экспрессирующие hpGUS[G:U], продуцировали значительно меньшее, но однородное количество антисмысловых кРНК. Наблюдение того, что растения с сильным сайленсингом, экспрессирующие hpGUS[G:U], накапливали намного более низкие уровни кРНК, чем растения с сильным сайленсингом, экспрессирующие hpGUS[wt], было любопытным и позволило авторам изобретения сделать предположение, что hpGUS[wt] процессируется по другому механизму в растениях, но все же обладал примерно такой же эффективностью, как и конструкция hpGUS[wt]. Дальнейшее наблюдение в этом эксперименте навело на то, что две относительно слабые антисмысловые полосы для растений hpGUS[G:U], по-видимому, имели такую же подвижность, как вторая и четвертая полосы, наблюдаемые для антисмысловых полос кРНК от hpGUS[wt]. Это было подтверждено в последующих экспериментах, описанных ниже. Авторы изобретения предположили, что четыре полосы для кРНК из hpGUS[wt] представляют 24-, 22-, 21- и 20-меры, и что РНК hpGUS[G:U] процессировалась главным образом с получением 22- и 20-мерных антисмысловых рРНК.
Важный, определенный вывод из данных, описанных выше, состоял в том, что молекула РНК hpGUS[G:U] процессировалась одним или несколькими ферментами Dicer с получением кРНК, в частности продукции антисмысловых кРНК, которые считаются медиаторами РНК-интерференции в присутствии различных белков, таких как Argonaute. Наблюдаемая продукция антисмысловых кРНК указывала, что также образовывались смысловые кРНК, но в экспериментах не определяли различия между деградацией/нестабильностью смысловых кРНК или неэффективным обнаружением смысловых кРНК из-за недостаточной гибридизации с зондом, который использовался. В результате этих экспериментов авторы изобретения также пришли к выводу, что в процессинге молекул РНК hpGUS[wt] и hpGUS[G:U] имеются четкие различия. Это указывало на то, что молекулы по-разному распознавались одной или несколькими рибонуклеазами Dicer.
Пример 8. Анализ кРНК из трансгенных растений, экспрессирующих модифицированные шпилечные РНК
Другой эксперимент по Нозерн-блот гибридизации проводили для обнаружения антисмысловых кРНК из hpGUS[G:U] растений и сравнения их размеров с размерами кРНК, полученных из hpGUS[wt]. Авторадиограф показан на Фигуре 18. В этот раз различие в размере двух полос антисмысловой кРНК из hpGUS[G:U] по сравнению с двумя основными полосами из hpGUS[wt] было более выраженным. Это лучше всего было заметно при сравнении подвижности полос в соседних дорожках 9 и 10 на Фигуре 18. Этот результат подтвердил, что две шпилечные РНК процессировались по-разному одной или несколькими рибонуклеазами Dicer в растениях.
Для дальнейшего изучения этого, небольшие популяции РНК из hpGUS[wt] и hpGUS[G:U] исследовали путем глубокого секвенирования суммарных линкер-амплифицируемых кРНК, выделенных из растений. Определяли частоту кРНК, которые были картированы в двухцепочечных областях шпилечной РНК. Также определяли распределение длины таких кРНК. Результаты показали увеличение частоты 22-мерных антисмысловых РНК из конструкции hpGUS[G:U] по сравнению с конструкцией hpGUS[wt]. Увеличение доли кРНК длиной 22 нт указывает на сдвиг в процессинге шпилечной hpGUS[G:U] рибонуклеазой Dicer-2 по сравнению с hpGUS[wt].
Пример 9. Анализ метилирования ДНК трансгенов в растениях
Наблюдения относительно вариабельности степени сайленсинга GUS, вызванного hpGUS[wt], и того, что антисмысловые 24-мерные кРНК были обнаружены в растениях hpGUS[wt], но, по-видимому, не в растениях hpGUS[G:U], привело авторов изобретения к вопросу, различались ли эти две популяции растений по уровню метилирования ДНК гена-мишени GUS. Считается, что сиквенс-специфические 24-мерные кРНК участвуют в стимуляции метилирования ДНК структур инвертированных повторов в растениях (Dong et al., 2011). Поэтому авторы изобретения исследовали уровни метилирования ДНК трансгена GUS в растениях hpGUS, в частности, в области 35S промотора гена, кодирующего шпильку (гена сайленсинга).
Для этого использовали зависимую от метилирования ДНК эндонуклеазу McrBC. McrBC является доступной в продаже эндонуклеазой, которая расщепляет ДНК, содержащую основания метилцитозина (mC), на одной или обеих цепях двухцепочечной ДНК (Stewart et al., 2000). McrBC распознает сайты на ДНК, которые состоят из двух полусайтов формы 5'-(G или A)mC-3', предпочтительно GmC. Эти полусайты могут быть разделены несколькими сотнями пар оснований, но оптимальный интервал составляет от 55 до приблизительно 100 пн. Двухцепочечная ДНК, содержащая такие связанные динуклеотиды GmC на обеих цепях, служит в качестве лучшего субстрата. Активность McrBC зависит от метилирования одного или обоих GC динуклеотидов. Поскольку растительная ДНК может быть метилирована по C в последовательностях CG, CHG или CHH, где H означает A, C или T (Zhang et al., 2018), расщепление ДНК при использовании McrBC с последующей ПЦР-амплификацией ген-специфических последовательностей может использоваться для обнаружения присутствия или отсутствия mC в специфических последовательностях ДНК в геномах растений. В этом анализе ПЦР-амплификация расщепленной McrBC геномной ДНК, которая метилирована, дает сниженные количества продукта амплификации по сравнению с ДНК, которая не метилирована, но будет давать такое же количество ПЦР-продукта, что и необработанная ДНК, если ДНК не метилирована.
Геномную ДНК выделяли с помощью стандартных методов из растений, содержащих конструкцию hpGUS[wt], hpGUS[G:U] или hpGUS[1:4] в дополнение к гену-мишени GUS (Draper and Scott, 1988). Очищенные образцы ДНК обрабатывали McrBC (номер по каталогу M0272; New England Biolabs, Massachusetts) согласно инструкциям производителя, включая присутствие иона Mg2+ и ГТФ, необходимых для эндонуклеазной активности. Вкратце, приблизительно 1 мкг геномной ДНК расщепляли McrBC в течение ночи в объеме реакции 30 мкл. Расщепленные образцы ДНК разбавляли до 100 мкл и представляющие интерес области ПЦР-амплифицировали следующим образом.
Обработанные образцы ДНК использовали в реакциях ПЦР с использованием следующих праймеров. Для последовательности соединения 35S-GUS для hpGUS[wt]: Прямой праймер (35S-F3), 5'-TGGCTCCTACAAATGCCATC-3' (SEQ ID NO:60); Обратный праймер (GUSwt-R2), 5'-CARRAACTRTTCRCCCTTCAC-3' (SEQ ID NO:61). Для последовательности соединения 35S-GUS для hpGUS[G:U]: Прямой праймер (GUSgu-R2), 5'-CAAAAACTATTCACCCTTCAC-3' (SEQ ID NO:62); обратный праймер (GUS4m-R2), CACRAARTRTACRCRCTTRAC (SEQ ID NO:63). Для последовательности промотора 35S для обеих конструкций: Прямой праймер (35-F2), 5'-GAGGATCTAACAGAACTCGC-3' (SEQ ID NO:64); обратный праймер (35-R1), 5'-CTCTCCAAATGAAATGAACTTCC-3' (SEQ ID NO:65). В каждом случае R=A или G, Y=C или T. Реакции ПЦР проводили со следующими параметрами циклов: 94°C - 1 мин, 35 циклов при 94°C - 30 сек, отжиг при 55°C - 45 сек, достройка при 68°C - 1 мин и оклюнчательная достройка при 68°C - 5 мин. Продукты ПЦР-амплификации подвергали электрофорезу и количественно определяли интенсивность полос.
Репрезентативные результаты показаны на Фигурах 19 и 20. Для области соединения 35S-GUS, которая включала 200 пн последовательности 35S промотора, включающую сайт инициации транскрипции, большинство растений hpGUS[wt] показали значительные уровни метилирования ДНК. Внутри популяции растений hpGUS[wt] отдельные растения, которые сохраняли более высокие уровни активности GUS, то есть с менее сильным сайленсингом, по-видимому, имеют более высокий уровень метилирования промоторной-GUS смысловой соединенной области. Результаты были аналогичными для области 35S промотора. С другой стороны, большинство растений hpGUS[G:U] и hpGUS[1:4] показали более слабое метилирование ДНК в области соединения 35S-GUS. Авторы изобретения предположили, что эта проксимальная промоторная последовательность важна для экспрессии трансгена, и метилирование в этой области, вероятно, будет снижать экспрессию конструкции сайленсинга посредством транскрипционного сайленсинга (TGS) трансгена. Это называется "аутосайленсинг".
Общее обсуждение касательно Примеров 6-9
Прерывание структуры инвертированных повторов ДНК в трансгене повышает его стабильность
Обе популяции трансгенных растений hpGUS[wt] и hpGUS[2:10] продемонстрировали широкий диапазон степени сайленсинга гена-мишени. С другой стороны, обе популяции, содержащие hpGUS[G:U] и hpGUS[1:4] растения, демонстрировали относительно единообразный сайленсинг GUS во многих независимых линиях, с сильным сайленсингом, наблюдаемым в случае первой конструкции, и относительно более слабым, но все же существенным снижением активности гена под действием последней конструкции. В шпилечных РНК из конструкций [G:U] и [1:4] около 25% нуклеотидов в смысловой и антисмысловой последовательностях были либо включены в пары оснований G:U, либо в некомплементарные основания в последовательности, которые были равномерно распределены по всему протяжению 200-нуклеотидных смысловых/антисмысловых последовательностей. Из-за дивергенции последовательностей между смысловой и антисмысловой последовательностями предположили, что некомплементарные основания в конструкциях ДНК между смысловым и антисмысловым "плечами" или инвертированной запрашиваемой структурой значительно нарушают такую структуру ДНК с инвертированными повторами. Повторяющиеся структуры ДНК могут привлекать метилирование ДНК и сайленсинг в различных организмах (Hsieh and Fire, 2000). Конструкция hpGUS[2:10] также включала некомплементарные основания между смысловой и антисмысловой областями, но при этом каждое 2 пн некомплементарное основание между смысловой и антисмысловой последовательностями было фланкировано последовательными 8 пн сопадающими комплементарными основаниями, поэтому некомплементарные основания могли не нарушать структуру инвертированных повторов ДНК настолько, как в трансгенах [G:U] и [1:4]. Таким образом, единообразие сайленсинга GUS, индуцированного hpGUS[G:U] и hpRNA[1:4], могла быть обусловлена, по меньшей мере частично, прерыванием структуры инвертированных повторов ДНК, что приводило к уменьшению метилирования и, следовательно, к снижению аутосайленсинга двух трансгенов. Другое преимущество некомплементарных оснований между областями смысловой и антисмысловой ДНК состояло в том, что клонирование инвертированного повтора в E. coli было улучшено, поскольку бактерии обычно удаляют или перестраивают совершенные инвертированные повторы.
Термодинамическая стабильность шРНК важна для степени сайленсинга гена-мишени
При сравнении трансгенных линий только с сильным сайленсингом, растения hpGUS[wt] имели наибольшую степень даунрегуляции гена-мишени, затем в указанном порядке следовали hpGUS[G:U], hpGUS[2:10] и hpGUS[1:4]. Анализ RNAFold предсказал, что структура шпильки РНК hpGUS[wt] имела самую низкую свободную энергию, т.е. наибольшую стабильность, затем следовали шпильки hpGUS[G:U], hpGUS[2:10] и hpGUS[1:4]. Авторы изобретения предположили, что чем стабильней структура шпильки РНК, тем больше степень сайленсинга гена-мишени, который она может индуцировать. Это также было в пользу более длинных двухцепочечных структур РНК, чем более коротких. Формирование стабильной двухцепочечной РНК, как предположили, было необходимо для эффективного процессинга Dicer. Результаты экспериментов, описанных в настоящем документе, указали на другое важное преимущество спаренной конструкции G:U по сравнению с конструкциям, включающим главным образом простые некомплементарные нуклеотиды, такие как hpGUS[1:4]; тогда как оба типа конструкций содержали прерванные структуры инвертированных повторов ДНК, что уменьшало аутосайленсинг на уровне РНК, РНК hpGUS[G:U] была более стабильной из-за способности G и U образовывать пары оснований. Также считали полезной комбинацию двух типов модификаций, включающую пары оснований G:U и включающую некоторые неспаренные нуклеотиды в двухцепочечной структуре РНК, но с относительно более высоким числом нуклеотидов, включенных в пары оснований G:U, чем в некомплементарных основаниях, по меньшей мере в 2, 3, 4 или даже 5 раз.
РНК hpGUS[G:U] эффективно подвергалась процессингу Dicer
Один важный вопрос, на который был дан ответ в этих экспериментах, состоял в том, может ли содержащая некомплементарные основания или G:U-спаренная шРНК процессироваться Dicer с образованием коротких РНК (кРНК). Сильный сайленсинг в растениях hpGUS[G:U] и в растениях 1:4 и 2:10 шРНК, содержащей некомплементарные основания, означал, что эти шпилечные РНК-структуры процессировались Dicer. Это было подтверждено для молекулы [G:U] с помощью кРНК нозерн-блот гибридизации, которая легко обнаруживала антисмысловые кРНК. Кроме того, степень сайленсинга GUS в растениях hpGUS[G:U] показала хорошую корреляцию с количеством накопленных антисмысловых кРНК. Анализ малых РНК методом глубокого секвенирования из двух выбранных линий из каждой (только одной для hpGUS[wt]) подтвердил, что растения hpGUS[G:U], такие как растения hpGUS[wt], генерировали большое количество кРНК, тогда как растения hpGUS[1:4] также генерировали кРНК, но с гораздо меньшей представленностью (Фигура 21). Более низкий уровень кРНК в растениях hpGUS[1:4] соответствовал относительно низкой эффективности сайленсинга GUS и указывал, что низкая термодинамическая стабильность стебля дцРНК в РНК hpGUS[1:4] снижала эффективность процессинга Dicer. Было отмечено, что степень сайленсинга GUS демонстрирует относительно слабую корреляцию с уровнем кРНК для конструкции hpGUS[wt], причем некоторые линии с сильным сайленсингом содержали относительно низкие количества кРНК. Это говорит о том, что сайленсинг GUS в некоторых линиях hpGUS[wt] был связан, по меньшей мере частично, с сайленсингом транскрипции, а не с кРНК-направленным PTGS. Авторы изобретения обнаружили, что аутосайленсинг гена, кодирующего шпильку, включавшего метилирование последовательностей генов, таких как область промотора, был снижен в случае использования модифицированных конструкций шпилечной РНК, в частности конструкции G:U.
G:U и 1:4 шРНК трансгены показали уменьшенное метилирование ДНК в проксимальной области 35S промотора
ПЦР-анализ перевара McrBC показал, что уровни метилирования ДНК в 240 пн последовательностях 35S вблизи сайта инициации транскрипции (TSS) были снижены в трансгенных популяциях hpGUS[G:U] и hpGUS[1:4] по сравнению с популяцией hpGUS[wt]. Этот результат указал авторам изобретения, что прерывание совершенной структуры инвертированных повторов из-за модификаций C→T (в hpGUS[G:U]) или 25% некомплементарных нуклеотидов (в hpGUS[1:4]) в смысловой последовательности приводили к снижению до минимума транскрипционный аутосайленсинг трансгенов шРНК. Это согласовывалось с единообразием сайленсинга гена GUS, наблюдаемого в популяциях hpGUS[G:U] и hpGUS[1:4] в сравнении с популяцией hpGUS[wt]. Авторы изобретения установили, что конструкция hpGUS[G:U] была более совершенной, чем конструкция hpGUS[1:4] в снижении метилирования промотора и, следовательно, транскрипционного аутосайленсинга, по меньшей мере, потому что она содержала уменьшенное количество, или даже не содержала, цитозин-нуклеотиды в смысловой последовательности, и поэтому не привлекла метилирование ДНК, которое могло распространяться на промотор.
Пример 10. Дизайн и тестирование шпилечных РНК, включающих пары оснований G:U и направленно воздействующих на эндогенные гены
Модифицированные шпилечные РНК, направленно воздействующие на РНК EIN2 и CHS
Так как G:U модифицированная шпилечная РНК, по-видимому, индуцирует более однородный и единообразный сайленсинг гена-мишени по сравнению с обычной шпилечной РНК, как описано выше, авторы изобретения захотели проверить, будет ли улучшенная конструкция также уменьшать экспрессию эндогенных генов. Поэтому авторы изобретения разработали, получили и протестировали несколько [G:U]-модифицированных конструкций шпилечных РНК, направленных либо на ген EIN2 или CHS, либо на оба гена, которые были эндогенными генами в Arabidopsis thaliana, выбранными в качестве типичных генов-мишеней в попытке вызвать сайленсинг. Ген EIN2 (SEQ ID NO:19) кодирует этилен-нечувствительный белок 2 (EIN2), который является центральным фактором в сигнальных путях, регулируемых сигнальной молекулой растений, этиленом, то есть является регуляторным белком, и ген CHS (SEQ ID NO:20) кодирует фермент халконсинтазу (CHS), которая участвует в синтезе антоцианинов в семенной оболочке A. thaliana. Была получена другая G:U модифицированная конструкция, которая одновременно направленно воздействовала на гены EIN2 и CHS, в которой последовательности EIN2 и CHS были транскрипционно слиты с получением одной шпилечной РНК. Кроме того, были получены три дополнительные конструкции, направленные либо на EIN2 или CHS, либо и на EIN2, и на CHS, в которых цитидиновые основания как в смысловой, так и в антисмысловой последовательностях были заменены тимидиновыми основаниями (в настоящем документе обозначена как конструкция G:U/U:G), а не только в смысловой последовательности, что также было сделано для модифицированных шпилек, направленно воздействующих на GUS. Модифицированные конструкции шпилечных РНК тестировали на их способность снижать экспрессию эндогенного гена EIN2 или генов EIN2 и CHS при использовании метода доставки генов для введения шпилечных РНК в клетки. Обычные шпилечные РНК, используемые в качестве контроля в эксперименте, имели область двухцепочечной РНК длиной 200 пар оснований для направленного воздействия на мРНК EIN2 или CHS по отдельности, или химерную область двухцепочечной РНК, содержащую 200 пар оснований из каждого гена EIN2 и CHS, которые были слиты вместе в виде единой шпилечной молекулы. В слитой РНК двухцепочечная часть EIN2 примыкала к петле шпильки, а область CHS была дистальной по отношению к петле. Все пары оснований в двухцепочечной области контрольной шпилечной РНК были каноническими парами оснований.
Получение конструкции
Фрагменты ДНК, охватывающие 200 пн области EIN2 дикого типа (SEQ ID NO:19) и кДНК CHS (SEQ ID NO:20), амплифицировали с помощью ПЦР с кДНК Colid-0 Arabidopsis thaliana при использовании пар олигонуклеотидных праймеров EIN2wt-F (SEQ ID NO:66) и EIN2wt-R (SEQ ID NO:67) или CHSwt-F (SEQ ID NO:68) и CHSwt-R (SEQ ID NO:69) соответственно. Фрагменты встраивали в pGEMT-Easy, как для шпилечных конструкций GUS (Пример 6). Фрагменты ДНК, содержащие модифицированный смысловой фрагмент EIN2[G:U] (SEQ ID NO:22) и CHS[G:U] (SEQ ID NO:24), 200 пн модифицированный смысловой или 200 пн модифицированный антисмысловой EIN2[G:U] (SEQ ID NO:25) и модифицированные антисмысловые фрагменты CHS[G:U] (SEQ ID NO:26), каждый из которых фланкирован сайтами рестрикции, собирали при отжиге соответствующих пар олигонуклеотидов, EIN2gu-F+EIN2gu-R, CHSgu -F+CHSgu-R, asEIN2gu-F+asEIN2gu-R и asCHSgu-F+asCHSgu-R (SEQ ID NO:70-77) с последующей ПЦР достройкой 3'-концов с использованием Taq-полимеразы LongAmp. Все G:U-модифицированные фрагменты ПЦР клонировали в вектор pGEM-T Easy и предполагаемые нуклеотидные последовательности подтверждали секвенированием. Слитые фрагменты CHS[wt]::EIN2[wt], CHS[G:U]:EIN2[G:U] и asCHS[G:U]::asEIN2[G:U] получали при лигировании соответствующих фрагментов ДНК CHS и EIN2 по общему сайту XbaI в плазмиде pGEM-T Easy.
Кассеты 35S::смысловой фрагмент::интрон PDK::антисмысловой фрагмент::OCS-T получали аналогичным образом, как для конструкций hpGUS. По существу антисмысловые фрагменты вырезали из соответствующих плазмид pGEM-T Easy при расщеплении HindIII и BamHI и встраивали в pKannibal по сайтам BamHI и HindIII, при этом они находились в антисмысловой ориентации относительно 35S промотора. Затем смысловые фрагменты вырезали из соответствующей плазмиды pGEM-T Easy при использовании XhoI и KpnI и встраивали по тем же сайтам соответствующего клона, содержащего антисмысловую последовательсть. Все кассеты в плазмидах pGEM-T Easy вырезали NotI и встраивали в pART27 с получением конечных бинарных векторов для трансформации растений.
Выравнивания модифицированных смысловой[G:U] и антисмысловой[G:U] нуклеотидных последовательностей с соответствующими последовательностями дикого типа, на которых показаны положения замененных нуклеотидов, показаны на Фигурах 22-25. Дизайн кассет экспрессии для шпилечных РНК схематично показан на Фигуре 26.
Расчетную свободную энергию образования шпилечных РНК оценивали с помощью программы FOLD. Показатели были вычислены в (ккал/моль): hpEIN2[wt] -453,5; hpEIN2[G:U] -328,1; hpCHS [wt] -507,7; hpCHS[G:U] -328,5; hpEIN2[G:U/U:G] -173,5; hpCHS[G:Y/U:G] -186,0; hpCHS::EIN2[wt] -916,4; hpCHS::EIN2[G:U] -630,9; hpCHS::EIN2[G:U/U:G) -333,8.
Трансформация растений
Все EIN2, CHS и химерные EIN2/CHS конструкции использовали для трансформации растений Arabidopsis thaliana расы Col-0 с использованием метода погружения цветков (Clough and Bent, 1998). Для отбора трансгенных растений, семена, собранные с обработанных погружением цветков Agrobacterium, стерилизовали хлором и сеяли на среду MS, содержащую 50 мг/л канамицина. Несколько трансгенных линий получали для всех девяти конструкций (Таблица 1). Эти первичные трансформанты (поколение T1) переносили в почву, самоопыляли и выращивали до зрелости. Семена, собранные у этих растений (семена Т2), использовали для получения растений Т2 и проводили скрининг для поиска линий, которые были гомозиготными по трансгену. Их использовали для анализа сайленсинга EIN2 и CHS.
Таблица 1. Обзор трансгенных растений, полученных в генетическом фоне Col-0
Анализ степени сайленсинга EIN2
EIN2 является геном A. thaliana, кодирующим рецепторный белок, участвующий в реагировании на этилен. Ген экспрессируется в ростках вскоре после прорастания семян, а также позднее, при росте и развитии растения. Мутантная EIN2 рассада демонстрирует удлинение гипокотиля по сравнению с изогенной рассадой дикого типа при проращивании в темноте в присутствии 1-аминоциклопропан-1-карбоновой кислоты (ACC), промежуточного соединения в синтезе этилена в растениях. Поэтому экспрессию гена EIN2 и степень сайленсинга в трансгенных растениях анализировали путем проращивания семени на среде MS, содержащей 50 мкг/л ACC, в полной темноте и измерения длины их гипокотиля по сравнению с рассадой дикого типа. Длина гипокотиля является удобным фенотипом для легкого измерения и хорошим индикатором степени снижения экспрессии гена EIN2, указывающим на разные уровни сайленсинга EIN2. Растения с сайленсингом экспрессии гена EIN2, как ожидают, будут иметь различные степени удлинения гипокотиля в зависимости от уровня сайленсинга EIN2, примерно в диапазоне между рассадой дикого типа (короткие гипокотили) и нулевой мутантной рассадой (длинные гипокотили). Исследовали семена от 20 рандомизированно отобранных, независимо трансформированных растений для каждой конструкции. Семена от одного растения из 20 содержащих конструкцию hpCHS::EIN2[G:U], не проросли. Данные по длине гипокотиля показаны на Фигуре 27.
Линии hpEIN2[wt] показали значительный диапазон степени сайленсинга EIN2, причем 7 линий (линии растений 2, 5, 9, 10, 12, 14, 16 на Фигуре 27) четко показали низкие уровни сайленсинга или такую же длину гипокотиля, как у дикого типа, а остальные 13 линий имели средний или сильный сайленсинг EIN2. Отдельные растения в рамках каждой независимой линии демонстрировали диапазон степени сайленсинга EIN2, на что указывали различия в длине гипокотиля. Напротив, только две линии (линии растений 5, 18 на Фигуре 27), включающие конструкцию hpEIN2[G:U], демонстрировали слабый сайленсинг EIN2, а остальные 18 демонстрировали однородный сильный сайленсинг EIN2. Кроме того, отдельные растения в каждой из 18 линий, по-видимому, имели относительно однородный сильный EIN2 по сравнению с растениями, трансформированными конструкцией hpEIN2[wt]. Авторы изобретения пришли к выводу, что G:U модифицированная конструкция шпилечной РНК была способна вызывать более единообразный, менее вариабельный сайленсинг эндогенного гена, который был более единообразным и более предсказуемым, чем в случае обычной шпилечной РНК, направленной на ту же область эндогенной РНК.
Трансгенные популяции hpEIN2[wt] и hpEIN2[G:U] также различались по соотношению между степенью сайленсинга EIN2 и числом копий трансгена. Число копий трансгена было указано отношениями сегрегации по маркерному гену устойчивости к канамицину у растений потомства - отношение 3:1 устойчивых:чувствительных ростков, указывающее на одиночную вставку локуса, тогда как отношение, которое было намного выше, указывало на многолокусные вставки трансгена. Несколько многокопийных линий, трансформированных конструкцией hpEIN2[wt], показали низкий уровень сайленсинга EIN2, но это не относилось к линиям hpEIN2[G:U], где и однокопийные, и многокопийные линии показали сильный сайленсинг EIN2.
Ген EIN2 также показал сайленсинг в ростках, трансформированных слитой шпилечной РНК CHS::EIN2. Подобно растениям, содержащим одну конструкцию hpEIN2[G:U], рассада hpCHS::EIN2[G:U] четко продемонстрировала более единообразный сайленсинг EIN2 на независимых линиях, чем рассада hpCHS::EIN2 [wt]. Сайленсинг в отдельных растениях в рамках независимой линии также оказался более единообразным для линий hpCHS::EIN2[G:U], чем линий hpCHS::EIN2[wt]. В то же время степень сайленсинга EIN2 была немного сильнее для растений с высоким сайленсингом hpCHS::EIN2[wt], чем для растений hpCHS::EIN2[G:U], аналогично сравнению между растениями, трансформированными hpGUS[wt] и hpGUS[G:U]. Сравнение степени сайленсинга показало, что слитые конструкции не индуцируют более сильный сайленсинг EIN2, чем одиночная конструкция hpEIN2[G:U], фактически, слитая конструкция шпилечой G:U, по-видимому, индуцирует немного более слабый сайленсинг EIN2, чем одиночная конструкция hpEIN2[G:U], направленно воздействующая на ген.
При исследовании растений, трансформированных конструкциями G:U/U:G, в которых цитидин-нуклеотиды (C) как смысловой, так и антисмысловой последовательностей были модифицированы с заменой на тимидин-нуклеотиды (T), наблюдали незначительное увеличение или отсутствие увеличения длины гипокотиля для все 20 проанализированных независимых линий по сравнению с растениями дикого типа. Это наблюдали для hpEIN2[G:U/U:G] и для hpCHS::EIN2[G:U/U:G] конструкций. Эти результаты показали авторам изобретения, что G:U/U:G спаренные конструкции шпилечных РНК, имеющие около 46% замен, не были эффективными для индукции сайленсинга генов-мишеней, возможно, потому что спаривание шпилечных РНК было слишком сильно дестабилизировано. Авторы изобретения посчитали, что неэффективность может быть вызвана двумя возможными причинами. Во-первых, двухцепочечная область EIN2 шпилечных РНК содержала 92 пары оснований G:U из 200 потенциальных пар оснований между смысловой и антисмысловой последовательностями. Во-вторых, выравнивание модифицированной антисмысловой последовательности с комплементом смысловой последовательности дикого типа показало, что 49 замен C на T в антисмысловой последовательности могли снижать эффективность направленного взаимодействия антисмысловой последовательности с мРНК EIN2. По результатам этого эксперимента авторы изобретения пришли к выводу, что, по меньшей мере для гена-мишени EIN2, существует верхняя допустимая граница количества нуклеотидных замен, которые могут присутствовать в шпилечной РНК, и при этом она сможет сохранять достаточную эффективность для сайленсинга. Например, 92/200=46% замен, вероятно, было слишком высоким процентом.
Анализ степени сайленсинга CHS
Трансгенные растения исследовали по уровню экспрессии гена CHS с помощью количественной ПЦР с обратной транскрипцией (кОТ-ПЦР) на РНК, выделенной из цельных растений, выращенных in vitro на среде для культур тканей. Праймеры, используемые для мРНК CHS, были следующими: прямой праймер (CHS-200-F2), 5'-GACATGCCTGGTGCTGACTA-3' (SEQ ID NO:78); обратный праймер (CHS-200-R2) 5'-CCTTAGCGATACGGAGGACA-3' (SEQ ID NO:79). Праймеры, используемые для референсного гена актина 2, используемого в качестве стандарта, были следующими: прямой праймер (Actin2-For) 5'-TCCCTCAGCACATTCCAGCA-3' (SEQ ID NO:80) и обратный праймер (Actin2-Rev) 5'-GATCCCATTCATAAAACCCCAG-3' (SEQ ID NO:81).
Данные показали, что уровень мРНК CHS, накопленной в растениях, по сравнению с референсной мРНК гена актина 2 был снижен в пределах 50-96% (Фигура 28).
Семя A. thaliana, полностью лишенное активности CHS, имеет бледную семенную оболочку по сравнению с коричневым цветом семян дикого типа. Поэтому семя трансгенных растений исследовали визуально на цвет их семенной оболочки. Явное побледнение семенной оболочки наблюдали у семян нескольких растений, но не наблюдали у семян других растений, несмотря на снижение мРНК CHS в листьях тех растений. Предположили, впрочем, что фенотип цвета семенной оболочки проявлялся только тогда, когда активность CHS была почти полностью устранена в развивающейся оболочке семян во время роста растений. Кроме того, 35S промотор, возможно, не был достаточно активен в развивающейся семенной оболочке, чтобы обеспечивать уровень снижения активности CHS с возникновением фенотипа бледных семян, наблюдаемого у нулевых мутантов. Улучшение визуального цветового фенотипа семенной оболочки может быть достигнуто при использовании промотора, который является более активным в семенной оболочке.
Пример 11. Анализ кРНК из конструкций шпилечной РНК
Нозерн-блот гибридизацию проводили на образцах РНК для обнаружения антисмысловых кРНК из растений hpEIN2[G:U] и сравнения их количества и размеров с кРНК, полученными из hpEIN2[wt]. Этот зонд представлял собой 32P-меченный РНК-зонд, соответствующий смысловой последовательности из 200 нуклеотидов в конструкции hpEIN2[wt], гибридизацию проводили в условиях низкой жесткости для обеспечения возможности обнаружения более коротких (20-24 нуклеотидов) последовательностей. Авторадиограф меченного зондами Нозерн-блота показан на Фигуре 29. Этот эксперимент показал, что шпилечная РНК hpEIN2 [G:U] процессировалась в кРНК, и уровень накопления был относительно однородным у 9 проанализированных независимо трансформированных растений hpEIN2[G:U] по сравнению с линиями hpEIN2[wt]. Как и в аналогичном эксперименте для шпилечных РНК к GUS, различие в размере двух антисмысловых полос кРНК из hpEIN2[G:U] по сравнению с двумя основными полосами из hpEIN2[wt] было совершенно очевидным. Это лучше всего видно при сравнении подвижности полос в соседних дорожках 10 и 11 на Фигуре 28.
Для дальнейшего исследования, популяции коротких РНК из hpEIN2[wt] и hpEIN2[G:U] анализировали путем глубокого секвенирования совокупных популяций кРНК, выделенных из цельных растений. Определяли долю каждой популяции, которую картировали в двухцепочечных областях hpEIN2[wt] и hpEIN2[G:U]. Примерно из 16 миллионов считываний в каждой популяции около 50000 кРНК были картированы в двухцепочечной области hpEIN2[wt], тогда как только около 700 - в hpEIN2[G:U]. Это указывало на то, что из шпильки [G:U] образовывалось гораздо меньше кРНК. Также наблюдали увеличение доли EIN2-специфических 22-меров.
Пример 12. Анализ метилирования ДНК растений с сайленсингом EIN2
Результаты сайленсинга GUS и EIN2 показали, что конструкции шРНК, имеющие немодифицированные смысловые последовательности, индуцировали сильно различающиеся уровни сайленсинга гена-мишени по сравнению с конструкциями, имеющими модифицированные смысловые последовательности, обеспечивающие пары оснований G:U. Как описано выше, промоторная область конструкции hpGUS[G:U], по-видимому, имеет меньшее метилирование по сравнению с конструкцией hpGUS[wt]. Для проверки метилирования ДНК и сравнения трансгенных растений hpEIN2[wt] и hpEIN2[G:U], 12 растений из каждой популяции проанализировали на метилирование ДНК в 35S промоторе и в области соединения 35S-промотор-смыслового EIN2 с использованием метода McrBC. Праймеры, используемые для области 35S промотора: прямой праймер (Top-35S-F2), 5'-AGAAAATYTTYGTYAAYATGGTGG-3' (SEQ ID NO:82), обратный праймер (Top-35S-R2), 5'-TCARTRRARATRTCACATCAATCC-3' (SEQ ID NO:83). Праймеры, используемые для области соединения 35S промотора со смысловым EIN2: прямой праймер (Link-35S-F2), 5'-YYATYATTGYGATAAAGGAAAGG-3' (SEQ ID NO:84) и обратный праймер (Link-EIN2-R2), 5' -TAATTRCCACCAARTCATACCC-3' (SEQ ID NO:85). В каждой из этих последовательностей праймеров Y=C или T и R=A или G.
Количественную оценку степени метилирования ДНК производили путем проведения ПЦР-анализа в реальном времени. Для каждого растения вычисляли показатель: скорость амплификации фрагмента ДНК после обработки геномной ДНК McrBC/скорость амплификации фрагмента ДНК без обработки геномной ДНК McrBC.
Почти каждое растение hpEIN2[wt] показало значимые уровни метилирования ДНК 35S промотора, особенно в соединении 35S-EIN2, но некоторые растения показали более высокие уровни, чем другие. Как показано на Фигурах 30 и 31, все линии растений, представленные на дорожках 1, 4, 7, 9, 11 и 12, демонстрировали сильный сайленсинг EIN2, на что указывала большая длина гипокотиля. С другой стороны, остальные шесть линий, представленные на дорожках 2, 3, 5, 6, 8 и 10, демонстрировали относительно слабый сайленсинг EIN2, что приводило к более коротким гипокотилям. Эти линии с более слабым сайленсингом показали более сильное метилирование ДНК в промоторной и соединительной последовательностях, на что указывает гораздо более низкая интенсивность полосы ПЦР, когда геномную ДНК предварительно расщепляли McrBC. Количественные анализы ПЦР в реальном времени (кПЦР) подтвердили эти наблюдения (Фигура 31). Все 12 протестированных линий показали некоторую степень метилирования ДНК как в области 35S промотора, так и в области 35S смыслового соединения ("соединение"). Наибольшая степень метилирования, т.е. самый низкий показатель в анализах кПЦР, была отмечена для линий hpEIN2[wt] 2, 3, 5, 6, 8 и 10, что коррелирует с уменьшенной степенью сайленсинга, измеренной по длине гипокотиля. Эти результаты подтвердили, что снижение сайленсинга EIN2 в некоторых линиях hpEIN2[wt] было связано с повышенным метилированием промотора. Даже в линиях растений hpEIN2[wt] с сайленсингом EIN2 все еще наблюдались значимые уровни метилирования ДНК, особенно в области фрагмента соединения 35S-смыслового EIN2. В случае метилирования промоторов считается, что это вызывает транскрипционный сайленсинг. В случае сайленсирующих конструкций, как в данном случае, это является формой "аутосайленсинга".
В отличие от линий hpEIN2[wt], линии hpEIN2[G:U] показали меньшее метилирование ДНК как 35S промотора, так и соединения 35S-EIN2. Фактически, четыре из этих 12 линий G:U, соответствующих дорожкам 1, 2, 3 и 7 на Фигуре 30 (дорожки 13, 14, 15 и 20 на Фигуре 31), не имели явного метилирования ДНК, о чем свидетельствует одинаковая сила ПЦР-полосы McrBC-обработанных и необработанных образцов. При количественном анализе этих амплификаций с помощью кПЦР, шесть из 12 линий показали малое или полное отсутствие снижения фрагмента после обработки McrBC и, следовательно, малое или полное отсутствие метилирования ДНК, см. нижнюю панель на Фигуре 31, линии 13, 14, 15, 18, 19 и 20. Эти результаты показали, что относительно единообразный сайленсинг EIN2 конструкцией hpEIN2[G:U], по меньшей мере, в некоторых линиях, было обусловлен значительно меньшим метилированием промотора и, следовательно, меньшим транскрипционным аутосайленсингом по сравнению с hpEIN2[wt].
Эти выводы дополнительно подтверждали анализом геномной ДНК из трансгенных линий растений с секвенированием после обработки бисульфитом. В этом анализе использовали тот факт, что обработка ДНК бисульфитом превращала неметилированные цитозиновые основания в ДНК в урацил (U), но оставляла 5-метилцитозиновые основания (mC) без изменений. После обработки бисульфитом определенный интересующий участок ДНК амплифицировали в реакциях ПЦР таким образом, что амплифицировалась только смысловая цепь обработанной ДНК. Затем продукт ПЦР подвергали массовому секвенированию, определяя положения и степень метилирования отдельных цитозиновых оснований в сегменте ДНК. Таким образом, анализ предоставил информацию с разрешением один нуклеотид о статусе метилирования сегмента ДНК.
Три линии растений, показавшие наиболее сильные уровни сайленсинга EIN2 для каждого из hpEIN2[wt] и hpEIN2[G:U], анализировали путем секвенирования с обработкой бисульфитом, соответствующие линиям 1, 7 и 9 hpEIN2[wt] и линиям 13, 15 и 18 hpEIN2[G:U] на Фигуре 31. Эти линии растений показали наибольшую длину гипокотиля и поэтому, как ожидалось, имели наиболее низкие уровни метилирования ДНК из 20 линий для каждой конструкции. Результаты представлены на Фигурах 32 и 33 для hpEIN2[wt] и hpEIN2[G:U] соответственно. При сравнении было ясно, что многочисленные цитозины в области 35S промотора и смысловой области EIN2 в растениях hpEIN2[wt] были сильно метилированы. В отличие от этого, три линии растений hpEIN2[G:U] показали гораздо более низкие уровни метилирования цитозина в области 35S промотора.
Пример 13. Уровни метилирования ДНК в промоторе конструкции hpGUS[1:4]
При анализе геномной ДНК, выделенной из растений hpGUS[1:4], на метилирование ДНК с использованием McrBC и бисульфитного метода, как описано выше, аналогично наблюдали, что метилирование цитозиновых оснований в 35S промоторе и областях 35S промотор-GUS смысловой последовательности было меньше по сравнению с растениями hpGUS[wt].
Общее обсуждение касательно Примеров 10-13
Двухцепочечная РНК, содержащая пары оснований G:U, вызывает больше единообразный сайленсинг гена, чем обычная дцРНК
Аналогично конструкциям GUS, hpEIN2[G:U] и hpCHS:EIN2[G:U] индуцировали более однородный и единообразный сайленсинг EIN2, чем соответствующие конструкции hpRNA[wt], кодирующие обычную шпилечную РНК. Единообразие наблюдали не только по многим независимым трансгенным линиям, но также по родственным растениям внутри трансгенной линии, каждая из которых имеет одинаковую вставку трансгена. В дополнение к единообразию степень сайленсинга EIN2, индуцированного hpEIN2[G:U], была близка таковой у линий hpEIN2[wt] с сильным сайленсингом. Анализ сайленсинга гена CHS показал, что конструкция hpCHS[G:U] была эффективна при снижении уровней мРНК CHS на 50-97%, но лишь немногие растения демонстрировали четко видимый фенотип при побледнении семенной оболочки. Вероятное объяснение отсутствию видимых фенотипов окраски семян, заключается в том, что даже низкие уровни активности CHS могут быть достаточными для продукции флавоноидных пигментов. Другие возможные объяснения состояли в том, что 35S промотор не был достаточно активен при развитии семенной оболочке с образованием фенотипа, или что последовательность конструкции hpCHS[G:U] содержала 65 замен цитозина (32,5%), по сравнению только с 43 (21,5%) для последовательности EIN2 и 52 (26%) для последовательности GUS. Кроме того, многие из этих цитозиновых оснований в последовательности CHS встречаются в наборах из двух или трех последовательных цитозинов, поэтому не все они должны быть заменены. При замене всех цитозинов в смысловой цепи, это приводило к большему количеству пар оснований G:U в РНК hpCHS[G:U], чем в РНК hpEIN2[G:U] и hpGUS[G:U], возможно, большему, чем оптимальное. Чтобы убедиться в этом, другой набор конструкций CHS получали с использованием последовательности, содержащей ряд цитозиновых замен, примерно из 5%, 10%, 15%, 20% или 25% замещенных оснований цитозина. Эти конструкции тестировали и определяли оптимальный уровень.
Линии hpEIN2[G:U] экспрессируют более единообразные уровни миРНК
В соответствии с относительно единообразным сайленсингом гена EIN2, линии hpEIN2[G:U] накапливали кРНК с более единообразным уровнем в независимых линиях. Это подтвердило вывод, сделанный в отношении конструкций hpGUS, что [G:U] модифицированная шРНК эффективно процессировалась Dicer и была способна индуцировать эффективный сайленсинг гена-мишени.
Слитые конструкции также обеспечивают сайленсинг генов
Цель включения слитых конструкций CHS:EIN2 в эксперимент состояла в проверке, можно ли вызвать сайленсинг двух генов-мишеней с использованием одной конструкции, кодирующей шпильку. Эксперимент с GUS показал, что свободная энергия и, следовательно, стабильность шпилечной РНК положительно коррелировали со степенью сайленсинга гена-мишени. Результаты показали, что слитая конструкция CHS:EIN2 действительно приводила к сайленсингу обоих генов - для CHS, по меньшей мере, на уровне мРНК.
Две конструкции hpRNA, hpEIN2[G:U/U:G] и hpCHS:EIN2[G:U/U:G], в которых как смысловая, так и антисмысловая последовательности были модифицированы с заменой C на T, в результате чего 46% пар оснований были превращены из канонических пар оснований в пары оснований G:U, вызывали лишь слабый или не вызывали сайленсинг EIN2 в большей части трансгенных растений. Возможные объяснения включают в себя следующее: i) слишком много пар оснований G:U, что привело к неэффективному процессингу Dicer, и ii) кРНК, связывающаяся с мРНК-мишенью, включала слишком много пар оснований G:U, не вызывала эффективного расщепления мРНК, или комбинация факторов.
Увеличенное единообразие сайленсинга гена-мишени G:U спаренными конструкциями связано с снижением метилирования промотора
Анализ метилирования ДНК с помощью ПЦР с расщеплением McrBC и секвенирования с обработкой бисульфитом показал, что все линии растений hpEIN2[wt] демонстрировали метилирование ДНК в промоторной области, при этом степень метилирования отрицательно коррелировала с уровнем сайленсинга EIN2. Даже три наименее метилированных линии, при оценке по ПЦР с расщеплением McrBC, показали примерно 40% уровни метилирования ДНК в 35S промоторе относительно всех метилированных цитозинов. Широко распространенное метилирование промотора, как предположили, было обусловлено кРНК-направленным метилированием ДНК в повторяющейся последовательности EIN2, которое распространялось в смежную промоторную область. В отличие от линий растений шРНК[wt], ряд линий hpEIN2[G:U] показали низкое метилирование или отсутствие метилирования промотора, при этом большинство проанализированных растений показало менее метилированные цитозины. Как обсуждалось в отношении линий hpGUS, снижению метилирования могут способствовать несколько факторов: i) структура инвертированных повторов ДНК была прервана путем замены оснований C основаниями T в смысловой последовательности, и ii) смысловая последовательность EIN2 не содержала цитозинов, поэтому она не могла быть метилирована путем кРНК-направленного метилирования ДНК, и iii) сниженный уровень продукции 24-мерных РНК из-за изменения в структуре области дцРНК с парами оснований G:U, приводящий к изменениям в распознавании некоторыми Dicer-рибонуклеазами и в результате уменьшению активности Dicer 3 и/или Dicer 4 и относительно более высокой активности Dicer 2. Таким образом, трансген hpEIN2[G:U] может проявлять себя как нормальный не-РНКи трансген (такой как овер-экспрессируемый трансген), и метилирование промотора, наблюдаемое в некоторых линиях, было обусловлено профилями вставки T-ДНК, а не нативной структурой инвертированных повторов ДНК шРНК трансгена.
Пример 14. Модифицированные шпильки для снижения экспрессии другого эндогенного гена
Были разработаны и синтезированы генетические конструкции для получения модифицированных сайленсирующих РНК либо для шпилечных РНК, либо для кпРНК, направленно воздействующие на другие эндогенные гены. Они включали следующее.
Ген FANCM в A. thaliana и в Brassica napus кодирует белок группы комплементации M анемии Фанкони (FANCM), который представляет собой белок РНК-геликазу с DEAD/DEAH боксом, рег. номера NM_001333162 и XM_018659358. Нуклеотидная последовательность кодирующей белок области кДНК, соответствующей гену FANCM A. thaliana, представлена в SEQ ID NO:31, а для B. napus - в SEQ ID NO:32.
Генетические конструкции были сконструированы и получены для экспрессии шпилечных РНК с заменами C на T или без замен и кпРНК, направленно воздействующей на ген FANCM в A. thaliana и в Brassica napus. Область-мишень была выбрана в гене A. thaliana: нуклеотиды 675-1174 (500 нуклеотидов) SEQ ID NO:31. Область-мишень была выбрана в гене B. napus: нуклеотиды 896-1395 (500 п.н.) SEQ ID NO:32. Были сконструированы и собраны конструкции, кодирующие шпилечные РНК, в которых использовали смысловую последовательность дикого типа или модифицированную (G:U) смысловую последовательность. Нуклеотидные последовательности конструкций hpFANCM-At[wt], hpFANCM-At[G:U], hpFANCM-Bn[wt] и hpFANCM-Bn[G:U] представлены в SEQ ID NO:33-36. Для создания конструкций G:U все цитозиновые основания в смысловых последовательностях заменяли тиминовыми основаниями - 102/500 (с получением 20,4% пар оснований G:U) в конструкции A. thaliana и 109/500 (21,8% оснований G:U)) в конструкции B. napus. Наиболее длинный непрерывный участок из канонических пар оснований в двухцепочечной области G:U модифицированной шпильки B. napus составлял 17 пар оснований, а второй по длине - 16 последовательных пар оснований.
Ген DDM1 в B. napus кодирует метилтрансферазу, метилирующую цитозиновые основания в ДНК (Zhang et al., 2018). Нуклеотидная последовательность кодирующей белок области кДНК соответствует гену DDM1 B. napus в SEQ ID NO:37.
Генетические конструкции были сконструированы и получены для экспрессии шпилечных РНК с заменами C на T или без замен и кпРНК, направленно воздействующей на ген DDM1 в Brassica napus. Были выбраны две несмежные области-мишени гена B. napus: нуклеотиды 504-815 и 1885-2074 с последовательностью SEQ ID NO:37, которые непосредственно соединяли с получением химерной смысловой последовательности. Таким образом, общая длина смысловой последовательности составила 502 нуклеотида. Были сконструированы и собраны конструкции, кодирующие шпилечные РНК, в которых использовали смысловую последовательность дикого типа или модифицированную (G:U) смысловую последовательность. Нуклеотидные последовательности конструкций hpDDM1-Bn[wt] и hpDDM1-Bn[G:U] представлены в SEQ ID NO:38-39. Для получения G:U конструкции цитозины в смысловых последовательностях заменяли тиминами - 106/502 (21,1% пар оснований G:U) в конструкции B. napus. Наиболее длинный непрерывный участок из канонических пар оснований в двухцепочечной области G:U модифицированной шпильки составлял 20 пар оснований, а второй по длине - 15 последовательных пар оснований.
Для другой конструкции, направленно воздействующей на эндогенный ген, разработали генетическую конструкцию для экспрессии шпилечной РНК с 95 заменами C на T в смысловой последовательности из 104 C в смысловой последовательности из 350 нуклеотидов, с получением 95/350=27,1% G:U пар оснований в двухцепочечной области шпилечной РНК. То есть не все C в смысловой последовательности были заменены T. В частности, если в последовательности присутствовал фрагмент из 3, 4 или 5 последовательных C, только 1 или 2 из трех C, или только 2 или 3 из четырех C, или только 2, 3 или 4 из 5 последовательных C были заменены T. Это обеспечило более равномерное распределение пар оснований G:U в области двухцепочечной РНК. Наиболее длинный непрерывный участок из канонических пар оснований в двухцепочечной области составлял 15 пар оснований, а второй по длине - 13 последовательных пар оснований.
Была разработана другая конструкция, в которой одна или две пары оснований в каждом блоке из 4, 5, 6 или 7 нуклеотидов были модифицированы с заменами C на T или A на G. Если смысловая последовательность дикого типа содержала участок из 8 или более нуклеотидов, состоящих из T или G, один или более нуклеотидов были заменены либо в смысловой цепи с получением некомплементарного нуклеотида в этом блоке, либо замена C на T или A на G была сделана в антисмысловой цепи, чтобы исключить возникновения двухцепочечного участка из 8 или более последовательных канонических пар оснований в двухцепочечной области полученной в результате шпилечной РНК, транскрибируемой с конструкции.
Пример 15. Модифицированные шпильки для снижения экспрессии генов в клетках животных
Для проверки модифицированных сайленсирующих РНК в клетках животных, в G:U спаренной форме, в форме кпРНК или в комбинации двух модификаций, в следующих экспериментах в качестве модельного гена-мишени использовали ген, кодирующий усиленный зеленый флуоресцентный белок (EGFP). Нуклеотидная последовательность кодирующей области EGFP показана в SEQ ID NO:40. Была выбрана область-мишень из 460 нуклеотидов, соответствующая нуклеотидам 131-591 SEQ ID NO:40.
Была сконструирована и получена генетическая конструкция, обозначенная hpEGFP[wt], которая экспрессировала шпилечную РНК, включающую, в порядке от 5' к 3' относительно промотора для экспрессии, антисмысловую последовательность EGFP из 460 нуклеотидов, которая была полностью комплементарна соответствующей области (нуклеотиды 131-590) кодирующей области EGFP, последовательность петли из 312 нуклеотидов, частично полученную из кодирующей области GUS (соответствует нуклеотидам 802-1042 ORF GUS), и смысловую последовательность EGFP из 460 нуклеотидов, которая была идентична по последовательности нуклеотидам 131-590 кодирующей области EGFP. Последовательность ДНК, кодирующая шпилечную РНК hpEGFP[wt] (SEQ ID NO:41), включала сайт рестрикции NheI на 5'-конце и сайт SalI на 3'-конце для обеспечения клонирования в вектор pCI (Promega Corporation). Этот вектор подходит для экспериментов по трансфекции клеток млекопитающих и должен обеспечивать экспрессию с сильного промотора/энхансера ЦМВ. Конструкция также содержла последовательность промотора Т7, встроенную между сайтом NheI и началом антисмысловой последовательности для обеспечения транскрипции in vitro с получением шпилечной РНК с использованием РНК-полимеразы Т7. Кассету, кодирующую шпильку, встраивали по сайтам NheI-SalI в вектор экспрессии pCI, в результате чего область, кодирующая РНК, была функционально связана с промотором ЦМВ и поздней областью полиаденилирования/терминации транскрипции SV40.
Была сконструирована и получена соответствующая шпилечная конструкция, которая содержала 157 замен C на T в смысловой последовательности и не содержала замен в антисмысловой последовательности, обозначенная hpEGFP[G:U] (SEQ ID NO:42). Область-мишень в области, кодирующей EGFP, представляла собой нуклеотиды 131-590. Процент замен C на T и, следовательно, пар оснований G:U в стебле шпилечной РНК составлял 157/460=34,1%. Смысловая и антисмысловая последовательности имели идентичную длину, 460 нуклеотидов. В области сайленсинга генов длинных двухцепочечных РНК обычно стараются избегать из-за вероятности активации клеточного ответа, включая активацию интерферонами.
Конструкция кпРНК, обозначенная ledEGFP[wt], была сконструирована и получена для экспрессии кпРНК, содержащей, в порядке от 5' к 3' относительно промотора для экспрессии, антисмысловую последовательность EGFP из 228 нуклеотидов, которая была полностью комплементарна нуклеотидам 131-358 кодирующей последовательности EGFP, последовательность петли из 150 нуклеотидов, смысловая последовательность EGFP из 460 нуклеотидов, которая была идентична по последовательности нуклеотидам 131-590 кодирующей области EGFP (SEQ ID NO:40), последовательность петли из 144 нуклеотидов и антисмысловая последовательность из 232 нуклеотидов, которая полностью комплементарна нуклеотидам 359-590 кодирующей последовательности EGFP, фланкированная сайтами рестрикции NheI и SalI (SEQ ID NO:43). Таким образом, кодируемая кпРНК относится к типу, показанному на Фигуре 1А. Структура кпРНК при отжиге на себя путем спаривания оснований между одной смысловой и двумя антисмысловыми последовательностями содержала двухцепочечную область из 460 пар оснований, соответствующую области-мишени EGFP, при этом две антисмысловые последовательности не были непосредственно ковалентно связаны друг с другом, но имели "пропуск" или "ник" между концами, соответствующими нуклеотидам 358 и 359. Структура кпРНК была встроена в более протяженный РНК транскрипт, включающий 5'- и 3'-области, полученные из последовательностей в промоторе ЦМВ и SV40-поздних областей полиаденилирования/терминации транскрипции.
Также была сконструирована и получена соответствующая конструкция кпРНК, которая содержала 162 замены C на T в смысловой последовательности и не содержала замен в антисмысловой последовательности, обозначенная ledEGFP[G:U] (SEQ ID NO:44). В каждом случае область-мишень в кодирующей области EGFP являлась нуклеотидами 131-590 относительно кодирующей белок области, начиная со старт-кодона ATG (SEQ ID NO:40). Процент замен C на T и, таким образом, пар оснований G:U в стебле кпРНК составлял 162/460=35,2%.
Плазмиды, кодирующие сайленсирующие РНК hpEGFP[wt], hpEGFP[G:U], ledEGFP[wt] и ledEGFP[G:U], тестировали на активность сайленсинга генов в клетках CHO, HeLa и VERO путем трансфекции векторов в клетки. Анализы проводили путем котрансфекции тестируемых плазмид с плазмидой, экспрессирующей GFP. Все анализы проводили в тройной повторности. Клетки СНО (клетки яичника китайского хомячка) и клетки VERO (клетки почек африканской зеленой мартышки) сеяли в 24-луночные планшеты при плотности 1×105 клеток на лунку. Клетки СНО выращивали в модификации MEMα (Sigma, USA), а клетки HeLa и VERO выращивали в DMEM (Invitrogen, USA). В обе базальных среды добавляли 10% эмбриональной бычьей сыворотки, 2 мМ глутамина, 10 мМ Hepes, 1,5 г/л бикарбоната натрия, 0,01% пенициллина и 0,01% стрептомицина. Клетки выращивали при 37°C в 5% CO2. Затем клетки трансфицировали 1 мкг на лунку плазмидной ДНК или миРНК в качестве контроля сайленсинга EGFP при использовании Lipofectamine 2000. Вкратце, тестируемую миРНК или плазмиду объединяли с репортерной плазмидой GFP (pGFP N1) и затем смешивали с 1 мкл Lipofectamine 2000, обе разбавляли в 50 мкл OPTI-MEM (Invitrogen, USA) и инкубировали при комнатной температуре в течение 20 мин. Затем комплекс добавляли к клеткам и инкубировали в течение 4 часов. Клеточную среду заменяли и инкубировали клетки в течение 72 часов. Затем клетки подвергали проточной цитометрии для измерения сайленсинга GFP. Вкратце, анализируемые клетки трипсинизировали, промывали в PBSA, ресуспендировали в 200 мкл 0,01% азида натрия и 2% FCS в PBSA и анализировали с использованием проточного цитофлуориметра FACScalibur (Becton Dickinson). Анализ данных проводили с использованием программы CELLQuest (Becton Dickinson) и представляли как среднюю интенсивность флуоресценции (MFI) в процентах от контрольных клеток с репортерной и нерелевантной (отрицательный контроль) шРНК.
миРНК против GFP, называемая si22, была получена от Qiagen (USA). Последовательность миРНК против GFP si22 была смысловой 5'-GCAAGCUGACCCUGAAGUUCAU-3' (SEQ ID NO:86) и антисмысловой 5'-GAACUUCAGGGUCAGCUUGCCG-3' (SEQ ID NO:87). Генетическую конструкцию положительного контроля, обозначенную pshGFP, создавали с помощью одностадийной реакции ПЦР при использовании мышиной последовательности U6 в качестве матрицы. В качестве прямого праймера использовали 5'-TTTTAGTATATGTGCTGCCG-3' (SEQ ID NO:88), и в качестве обратного праймера использовали 5'-CTCGAGTTCCAAAAAAGCTGACCCTGAAGTTCATCTCTCTTGAAGATGAACTTCAGGGTCAGCCAAACAAGGCTTTTCTCCAA-3' (SEQ ID NO:89). Продукт амплификации, который включал полноразмерную кассету экспрессии, лигировали в pGEM-T Easy. Нерелевантную контрольную плазмиду шРНК также конструировали с помощью того же метода ПЦР. Для ее конструирования использовали прямой праймер 5'-TTTTAGTATATGTGCTGCCG-3' (SEQ ID NO:90), и обратный праймер 5'-ctcgagttccaaaaaaataagtcgcagcagtacaatctcttgaattgtactgctgcgacttatgaataccgcttcctcctgag-3' (SEQ ID NO:91).
Полученные в результате одного эксперимента данные показаны на Фигуре 34. Четкое снижение активности EGFP (сайленсинг РНК) наблюдали как в клетках VERO, так и в клетках CHO, как для si22, так и для pshGFP-положительных контролей при сравнении с нерелевантным шРНК контролем. Эти положительные контроли являлись хорошо проверенной малой молекулой дцРНК (si22) или кодировали шРНК (pshGFP), которые, как известно, обладают очень сильной сайленсирующей активностью в клетках млекопитающих. Контрольные молекулы РНК имеют двухцепочечные области из 20 последовательных пар оснований и 21 последовательной пары оснований, соответственно, с использованием только канонических пар оснований и без некомплементарных нуклеотидов в двухцепочечных областях и в пределах 20-30 пар оснований, обычно используемых в клетках млекопитающих. С другой стороны, конструкции шРНК и кпРНК экспрессируют молекулы, имеющие протяженные области дцРНК. Было обнаружено, что все четыре конструкции специфично подавляют экспрессию EGFP в значимой степени в клетках обоих типов (Фигура 34). Включение замен G:U приводили к четкому улучшению сайленсинга для обеих конструкций в клетках СНО. В клетках VERO заметное улучшение сайленсинга наблюдали только при использовании конструкции ledEGFP[G:U] в сравнении с ledEGFP[wt].
Во втором эксперименте с использованием (человеческих) клеток HeLa и анализом активности EGFP через 48 ч после трансфекции, были получены аналогичные результаты (Фигура 35).
Важно отметить, что сайленсинг генов наблюдали в клетках млекопитающих при использовании эффекторных молекул шРНК и кпРНК, с учетом того, что они имели более протяженные двухцепочечные области, чем обычный диапазон от 20 до 30 пн. Также было ясно, что модификация с заменой нуклеотидов с образованием пар оснований G:U значительно усиливает эффект сайленсинга генов этих более длинных молекул дцРНК. Этот эффект может быть обусловлен тем, что эти структуры более схожи с эндогенными преРНК, предшественниками миРНК, наблюдаемыми в эукариотических клетках, и, таким образом, улучшают процессинг более протяженной дцРНК для загрузки в эффекторные белки РНК-индуцируемого комплекса сайленсинга (RISC).
Пример 16. Конструкции РНК, направленно воздействующие с генами DDM1 и FANCM в растениях
Авторы изобретения рассмотрели способы увеличения скорости, с которой могут быть получены и исследованы новые генетические профили и разнообразие (генетический эффект) для определения желаемых продуктивных признаков у растений. Один из способов, который был рассмотрен, заключался в том, чтобы найти способ увеличить скорость рекомбинации, которая происходит при половом размножении растений. Селекционеры растений полагаются на события рекомбинации для создания различных генетических (аллельных) комбинаций, среди которых они могут искать требуемый генетический профиль, связанный с повышением продуктивности. Однако количество событий рекомбинации на каждом этапе размножения чрезвычайно мало по сравнению с количеством возможных генетических профилей, которые можно исследовать. Кроме того, элементы, которые контролируют, где эти события происходят в геноме, исследованы недостаточно. Поэтому авторы изобретения рассмотрели вопрос о том, можно ли использовать кпРНК, доставляемую либо экзогенно, либо эндогенно посредством трансгенного подхода, для изменения скоростей рекомбинации у растений, чтобы обеспечить быстрое увеличение генетического разнообразия и сделать возможным более быстрое получение генетического эффекта в скрещиваемых популяциях.
Эпигеном растений находится под влиянием ряда различных химических модификаций ДНК и ассоциированных белков, которые организуют, упаковывают и стабилизируют геном. Эти модификации также регулируют, где происходит рекомбинация, при этом плотная упаковка генома является сильным ингибитором рекомбинации (Yelina et al., 2012; Melamed-Bessudo et al., 2012). Белок УМЕНЬШЕННОЕ МЕТИЛИРОВАНИЕ ДНК 1 (DDM1) представляет собой фермент, который регулирует метилирование ДНК и упаковку генома. Мутация в этом гене может изменять положение событий рекомбинации (Yelina et al., 2012; Melamed-Bessudo et al., 2012).
События рекомбинации во время мейоза жестко регулируются, при этом только 1-2 события происходят в каждой хромосоме, чтобы обеспечить надлежащую хромосомную сегрегацию в метафазе 1. События рекомбинации инициируются при образовании двухцепочечных разрывов (DSB) ДНК, которые создает фермент SPO11 (Wijnker et al., 2008). Это приводит к возникновению сотен DSB на хромосоме. Хотя некоторые из этих DSB приводят к кроссоверам, большинство восстанавливаются ферментами репарации ДНК, прежде чем может произойти событие рекомбинации. Кроме того, существует ряд негативных регуляторов, которые ингибируют превращение DSB в кроссоверы. В первоначальном подходе, предложенном авторами изобретения, генетические конструкции, кодирующие молекулы кпРНК или обычные молекулы шпилечных РНК для сравнения должны были вводить в растения A. thaliana с целью направленного воздействия на ген, кодирующий белковый фактор, который потенциально может влиять на скорость рекомбинации, такой как белок ГРУППЫ КОМПЛЕМЕНТАЦИИ M АНЕМИИ ФАНКОНИ (FANCM).
Нуклеотидная последовательность гена DDM1 A. thaliana была представлена под рег. номером AF143940 (Jeddeloh et al., 1999). Как было показано, снижение экспрессии гена DDM1 уменьшало метилирование ДНК и увеличивало число и количество положений событий кроссовера в A. thaliana (Melamed-Bessudo and Levy, 2012).
Brassica napus является аллотетраплоидным видом и имеет два гена DDM1 на каждом из субгеномов A и C, на хромосомах A7, A9, C7 и C9, и поэтому имеет в общей сложности четыре гена DDM1. Эти гены называются BnaA07g37430D-1, BnaC07g16550D-1, BnaA09g52610D-1 и BnaC09g07810D-1. Нуклеотидная последовательность гена DDM1 BnaA07g37430D-1 B. napus предоставлена под рег. номером XR_001278527 (SEQ ID NO:93). Была сконструирована и получена конструкция шпилечной РНК, направленно взаимодействующая с областью из 500 нуклеотидов четырех генов, соответствующая нуклеотидам 650-959 и 2029-2218 SEQ ID NO:93. Нуклеотидная область, используемая для конструирования конструкций шРНК и кпРНК, направленно воздействует на все четыре гена DDM1 BnaA07g37430D-1, BnaC07g16550D-1, BnaA09g52610D-1 и BnaC09g07810D-1, присутствующих в B. napus, на основе консервативности последовательности между генами. Порядок элементов в конструкции шРНК был следующим: промотор-смысловая последовательность-последовательность петли, включающая интрон из вектора Hellsgate-антисмысловая последовательность-терминатор транскрипции/область полиаденилирования. Нуклеотидная последовательность химерной ДНК, кодирующей шРНК, представлена в SEQ ID NO:94.
Была изготовлена вторая конструкция шпилечной РНК, кодирующая шпилечную РНК, направленно взаимодействующую с той же областью из 500 нуклеотидов и имеющая такую же структуру, за исключением того, что 97 цитозин-нуклеотидов (C) смысловой последовательности были заменены тимидин-нуклеотидами (T). При транскрипции химерной ДНК и отжиге на себя G:U замещенной шРНК происходило спаривание 97/500=19,4% нуклеотидов в области дцРНК с образованием пар оснований G:U. Нуклеотидная последовательность химерной ДНК, кодирующей G:U модифицированную шРНК, представлена в SEQ ID NO:95. Кроме того, была получена химерная ДНК, кодирующая кпРНК, направленно взаимодействующую с той же областью гена DDM1 B. napus. Нуклеотидная последовательность этой химерной ДНК, кодирующей кпРНК, представлена в SEQ ID NO:96.
Для получения РНК путем транскрипции in vitro препараты ДНК расщепляли рестриктазой HincII, которая создавала разрыв сразу после кодирующей области, транскрибировали in vitro с использованием РНК-полимеразы Т7, очищали РНК и затем концентрировали в водном буферном растворе. кпРНК использовали для направленного воздействия на эндогенные транскрипты DDM1 в семядолях B. napus (канола). Семядоли пятидневных ростков, выращенных в асептических условиях на среде для культур тканей, осторожно вырезали и помещали в чашку Петри, содержащую 2 мл жидкой среды MS, содержащей 2% (в/об) сахарозы, с 113 мкг кпРНК или 100 мкл водного буферного раствора в качестве контроля. Жидкая среда MS, используемая для обработок, содержала поверхностно-активное вещество Silwett-77 (0,5 мкл в 60 мл). Чашки Петри инкубировали в шейкере при легком встряхивании, чтобы семядоли пропитались раствором, содержащим кпРНК. Образцы собирали через 5 и 7 часов после нанесения кпРНК. В параллельном эксперименте верхнюю поверхность семядолей покрывали либо 10 мкг кпРНК, либо буферным раствором и инкубировали на влажной бумажной салфетке. Образцы собирали через 7 часов после нанесения кпРНК.
Кроме того, для направленного воздействия на эндогенные транскрипты DDM1 в репродуктивной ткани B. napus, цветочные почки канолы обрабатывали кпРНК либо в присутствии, либо в отсутствие аликвоты суспензии клеток штамма Agrobacterium tumafecians AGL1, то есть живых клеток AGL1. Водный буферный раствор с клетками AGL1 или без них служил в качестве соответствующего контроля. AGL1 выращивали в 10 мл жидкой среды LB, содержащей 25 мг/мл рифампицина, в течение двух дней при 28°C. Клетки собирали центрифугированием при 3000 об/мин в течение 5 минут. Осадок клеток промывали и клетки ресуспендировали в 2 мл жидкой среды MS. Цветочные почки инкубировали в чашке Петри, содержащей 2 мл жидкой среды MS, включающей 0,5 мкл Silwett-77, в 50 мл жидкой среды MS, с 62 мкг кпРНК или 62 мкг +50 мкл культуры AGL1. В качестве контроля использовали 50 мкл буферного раствора или 50 мкл буферного раствора +50 мкл культуры AGL1. Образцы инкубировали на шейкере с мягким встряхиванием в течение 7 часов. Три биологических повторности использовали для каждой обработки.
Обработанные и контрольные семядоли и цветочные почки два раза промывали в стерильной дистиллированной воде, с помощью бумажной салфетки удаляли воду с поверхности и быстро замораживали в жидком азоте. РНК выделяли из обработанных и контрольных тканей, обрабатывали ДНКазой для удаления геномной ДНК и подвергали количественному анализу. кДНК первой цепи синтезировали с использованием равных количеств суммарной РНК из образцов, обработанных кпРНК, и их соответствующих контролей. Экспрессию DDM1 анализировали с использованием количественной ПЦР в реальном времени (кОТ-ПЦР).
В обработанных семядолях, пропитанных кпРНК, представленность транскрипта DDM1 снижалась приблизительно на 83-86% через 5 ч, а затем еще падала со снижением 91% через 7 ч по сравнению с контролями. Аналогичным образом снижение уровня мРНК DDM1 по сравнению с контролем приблизительно на 78-85% наблюдали в семядолях, которые были покрыты кпРНК. В цветочных почках, обработанных кпРНК, не было обнаружено различий в представленности мРНК DDM1 по сравнению с контролем в отсутствие клеток Agrobacterium. Тем не менее, снижение приблизительно на 60-75% уровней транскрипта DDM1 наблюдали в цветочных почках, которые были обработаны кпРНК в присутствии Agrobacterium по сравнению с соответствующим контролем. Не было обнаружено существенного различия в уровнях транскрипта DDM1 при сравнении контроля без Agrobacterium с контролем, в котором присутствовала Agrobacterium, что показывало, что сами клетки Agrobacterium не вызывали снижения транскрипта DDM1. В совокупности эти результаты показали, что кпРНК способна снижать уровни эндогенного транскрипта DDM1 как в семядолях, так и в цветочных почках, тогда как живые клетки Agrobacterium, по-видимому, способствуют проникновению кпРНК в цветочные почки. Такая доступность кпРНК также может быть достигнута физическими способами, такими как прокалывание наружных слоев цветочных почек, центрифугирование или вакуумная инфильтрация, или с помощью комбинации таких способов.
У некоторых мутантов Arabidopsis thaliana, таких как мутанты zip4, отсутствуют мейотические кроссоверы, что вызывает нарушение сегрегации гомологов хромосом и, таким образом, снижает фертильность и приводит к укорачиванию стручков (плодов), которые можно визуально отличить от стручков растений дикого типа. Фенотип мутантов zip4 может быть устранен путем снижения экспрессии гена FANCM.
Нуклеотидная последовательность гена FANCM A. thaliana представлена под рег. номером NM_001333162 (SEQ ID NO:97). Была сконструирована и получена конструкция шпилечной РНК, направленно воздействующая на область гена из 500 нуклеотидов, соответствующую нуклеотидам 853-1352 SEQ ID NO:97. Порядок элементов в конструкции являлся следующим: промотор-смысловая последовательность-последовательность петли, включающая интрон из вектора Hellsgate-антисмысловая последовательность-терминатор транскрипции/область полиаденилирования. Нуклеотидная последовательность химерной ДНК, кодирующей шРНК, представлена в SEQ ID NO:98. Была получена вторая конструкция шпилечной РНК, кодирующая аналогичную шпилечную РНК, направленно воздействующую на ту же область из 500 нуклеотидов, за исключением того, что 102 цитозин-нуклеотидов (C) смысловой последовательности были заменены тимидин-нуклеотидами (T). При транскрипции химерной ДНК и отжиге на себя полученной G:U замещенной шРНК происходило спаривание 102/500=20,4% нуклеотидов в области дцРНК с образованием пар оснований G:U. Нуклеотидная последовательность химерной ДНК, кодирующей G:U-модифицированную шРНК, представлена в SEQ ID NO:99. Кроме того, была получена химерная ДНК, кодирующая кпРНК, последовательность на ту же область гена FANCM A. thaliana. Нуклеотидная последовательность этой химерной ДНК, кодирующей кпРНК, представлена в SEQ ID NO:100.
B. napus имеет по одному гену FANCM на каждом из его субгеномов A и C, обозначенные BnaA05g18180D-1 и BnaC05g27760D-1. Нуклеотидная последовательность одного из генов FANCM B. napus представлена под рег. номером XM_022719486.1; SEQ ID NO:101). Была сконструирована и получена химерная ДНК, кодирующая шпилечную РНК, направленно воздействующую на область гена из 503 нуклеотидов, соответствующую нуклеотидам 2847-3349 SEQ ID NO:101. Порядок элементов в конструкции являлся следующим: промотор-смысловая последовательность-последовательность петли, включающая интрон из вектора Hellsgate-антисмысловая последовательность-терминатор транскрипции/область полиаденилирования. Нуклеотидная последовательность химерной ДНК, кодирующей шРНК, представлена в SEQ ID NO:102. Была получена вторая конструкция шпилечной РНК, кодирующая аналогичную шпилечную РНК, направленно воздействующую на ту же область из 503 нуклеотидов, за исключением того, что 107 цитозин-нуклеотидов (C) смысловой последовательности были заменены тимидин-нуклеотидами (T). При транскрипции химерной ДНК и отжиге на себя полученной G:U замещенной шРНК происходило спаривание 107/500=21,4% нуклеотидов в области дцРНК с образованием пар оснований G:U. Нуклеотидная последовательность химерной ДНК, кодирующей G:U-модифицированную шРНК, представлена в SEQ ID NO:103. Кроме того, была получена химерная ДНК, кодирующая кпРНК, направленно воздействующую на ту же область гена FANCM B. napus. Нуклеотидная последовательность этой химерной ДНК, кодирующей кпРНК, представлена в SEQ ID NO:104.
Для продукции РНК путем транскрипции in vitro, препараты ДНК расщепляли рестриктазой HincII, которая создавала разрыв сразу после кодирующей области, транскрибировали in vitro с помощью РНК-полимеразы Т7, очищали РНК и затем концентрировали в водном буферном растворе. кпРНК использовали вместе с AGL1 Agrobacterium tumefacians для направленного воздействия на транскрипты FANCM в пре-мейотических почках мутанта zip4 A. thaliana. Стручки мутанта zip4 были более короткими, что можно легко наблюдать визуально, по сравнению со стручками дикого типа из-за ослабления образования кроссовера, что привело к снижению фертильности. Было показано, что репрессия FANCM у мутанта zip4 восстанавливает фертильность и восстанавливает длину стручков.
Соцветия A. thaliana zip4, содержащие пре-мейотические зародыши, обрабатывали кпРНК, направленно воздействующим на FANCM, вместе с AGL1 или буферным раствором с AGL1 в качестве контроля, в каждом случае в присутствии поверхностно-активного вещества, в данном случае Silwett-77. После завершения посева семян стручки, полученные из пре-мейотических почек, разрезали для определения количества семян. Из 15 стручков обработанных кпРНК образцов, два стручка содержали 10 семян, один стручок содержал 9 семян, тогда как количество семян в контрольных стручках изменялось в пределах 3-6. Эти результаты показали, что наблюдаемое увеличение количества семян было связано с репрессией уровней транскрипта FANCM под действием кпРНК, что приводит к увеличению числа мейотических кроссоверов и повышению фертильности.
Пример 17. Конструкции РНК для резистентности к грибковому заболеванию
кпРНК, направленно воздействующая на гены Mlo ячменя и пшеницы
Грибковое заболевание зерновых растений, мучнистая роса, которое вызвает аскомицет Blumeria graminis f. sp. hordei в ячмене и родственный гриб Blumeria graminis f. sp. tritici в пшенице. B. graminis является облигатным биотрофным грибковым патогенном из отряда Erysiphales (Glawe, 2008), которому для размножения требуется растение-хозяин, включая непосредственное взаимодействие между грибом и клетками-хозяевами, чтобы гриб мог получать питательные вещества из растения. Вначале гриб поражает эпидермальный слой листьев, листовых влагалищ или колосьев после контакта аскоспор или конидий гриба с поверхностью. Листья остаются зелеными и активными в течение некоторого времени после заражения, затем разрастаются порошистые, мицелиальные массы, листья постепенно становятся хлоротическими и отмирают. По мере развития заболевания мицелий гриба может быть усеян мелкими черными точками, которые являются половыми плодоносящими телами гриба. Такое заболевание, как мучнистая роса, распространено по всему миру и наиболее опасно в прохладном, влажном климате. Болезнь влияет на урожай зерна главным образом за счет уменьшения количества головок, а также уменьшения размера и веса зерна. В настоящее время борьба с болезнями осуществляется путем обработки сельскохозяйственных культур аэрозолями фунгицида, который необходимо часто применять в прохладных и влажных условиях, и является дорогостоящей, или необходимо выращивать устойчивые сорта. Кроме того, устойчивость к фунгицидам у мучнистой росы на пшенице развивалась в Австралии.
Гены Mlo ячменя и пшеницы кодируют полипептиды Mlo, которые придают восприимчивость к B. graminis по неизвестному механизму. Существует множество близко родственных белков MLO, кодируемых семейством генов Mlo, которые являются уникальными для растений. Каждый ген кодирует белок с семью трансмембранными доменами и неизвестной биохимической активностью, локализованный в плазматической мембране. Примечательно, что только специфические гены Mlo в семействе способны действовать как гены восприимчивости к мучнистой росе, и они кодируют полипептиды с консервативными мотивами в цитоплазматическом C-концевом домене белков Mlo. Механизм, с помощью которого полипептиды Mlo действуют как факторы восприимчивости к мучнистой росе, неизвестен. Появление природных mlo мутантов пшеницы не было описано, по-видимому, из-за полиплоидной природы пшеницы. Однако искусственно созданные мутанты mlo демонстрируют некоторую устойчивость к заболеванию, но часто демонстрируют существенно сниженную урожайность зерна или преждевременное старение листьев (Wang et al., 2014; Acevedo-Garcia et al., 2017).
Гексаплоидная пшеница имеет три гомолога генов Mlo, обозначенных TaMlo-A1, TaMlo-B1 и TaMlo-D1, которые расположены на хромосомах 5AL, 4BL и 4DL соответственно (Elliott et al., 2002). Нуклеотидные последовательности кДНК, соответствующие генам, доступны под рег. номерами: TaMlo-A1, AF361933 и AX063298; TaMlo-B1, AF361932, AX063294 и AF384145; и TaMlo-D1, AX063296. Нуклеотидные последовательности генов в геномах A, B и D и аминокислотные последовательности кодируемых полипептидов идентичны приблизительно на 95-97% и 98% соответственно. Все три гена экспрессируются в листьях растений, при этом уровни экспрессии повышаются по мере роста и созревания растений. Поэтому авторы изобретения разработали и получили конструкцию кпРНК, которая может снижать экспрессию всех трех генов, воспользовавшись степенью идентичности последовательностей между генами и направленно воздействуя на область гена с высокой степенью консервативности последовательности.
Была получена химерная ДНК, кодирующая конструкцию кпРНК, направленно воздействующую на все три гена TaMlo-A1, TaMlo-B1 и TaMlo-D1. Генетическая конструкция была получена с помощью принципов создания кпРНК, описанных выше, с разделенной последовательностью, являющейся антисмысловой последовательностью, и сплошной последовательностью, являющейся смысловой последовательностью (Фигура 1A).
Была выбрана нуклеотидная последовательность длиной 500 пн гена-мишени TaMlo, соответствующая нуклеотидам 916-1248, слитым с 1403-1569 SEQ ID NO:136. Область дцРНК каждой кпРНК имела длину 500 пн; смысловая последовательность в области дцРНК являлась непрерывной сплошной последовательностью, например, соответствовала нуклеотидам 916-1248, слитым с 1403-1569 SEQ ID NO:136. Нуклеотидная последовательность, кодирующая кпРНК, представлена в настоящем документе в SEQ ID NO:137.
кпРНК получали путем транскрипции in vitro при использовании РНК-полимеразы T7, очищали и ресуспендировали в буфере. По 10 мкг кпРНК на лист наносили кистью на зону листьев растений пшеницы на стадии роста 23 по шкале Задокса. В качестве контроля некоторые листья подвергали ложной обработке с использованием только буфера. Обработанные и контрольные образцы листьев собирали и выделяли РНК. Анализы кПЦР на выделенных РНК показали, что уровни мРНК TaMlo, являющиеся комбинацией трех мРНК TaMlo, были снижены на 95,7%. Растения на стадии роста Z73 также обрабатывали и анализировали. Они показали снижение экспрессии гена TaMlo на QPCR на 91% по сравнению с контрольными образцами листьев. Снижение экспрессии гена TaMlo, наблюдаемое в обработанных участках листьев, было специфичным для обработанных зон - в дистальных, необработанных частях листьев снижение уровней мРНК TaMlo отсутствовало.
В mlo мутантах ячменя наблюдается повышение экспрессии различных генов, связанных с защитой от заболеваний. Поэтому обработанные кпРНК листья пшеницы анализировали с помощью кПЦР на уровни связанных с защитой генов, кодирующих PR4, PR10, β-1,3-глюканазу, хитиназу, гермин и фактор рибозилирования АДФ. Уровень экспрессии ни у одного из этих генов не был значимо изменен в обработанных участках листьев по сравнению с контрольными участками листьев.
Для проверки способности кпРНК повышать устойчивость к заболеваниям путем снижения экспрессии гена Mlo споры гриба мучнистой росы наносят на обработанные и необработанные зоны листьев. Листья отделяли от растений пшеницы, обрабатывали кпРНК, как раньше, и выдерживали на среде (50 мг бензимидазола и 10 г агара на литр воды) для предотвращения старения листьев, при освещении. Через двадцать четыре часа листья инокулировали спорами мучнистой росы и наблюдали развитие заболевания в течение 5-24 дней. На обработанных листьях наблюдался слабый или полностью отсутствовал рост мицелия гриба и отсутствовал хлороз листьев по сравнению с контрольными листьями, не обработанными кпРНК, которые показали обширный рост мицелия, окруженного хлоротическими зонами.
В последующих экспериментах более низкие уровни кпРНК применяли для определения минимального уровня кпРНК, который является эффективным. Кроме того, листья инокулировали через 2, 4, 7 или более дней после обработки кпРНК, чтобы увидеть, как долго сохраняется защитный эффект. Целые растения также опрыскивали препаратами кпРНК и проверяли на устойчивость к болезням после инокуляции патогенным грибом.
кпРНК, направленно воздействующая на гены VvMLO Vitis vinifera
Гены MLO Vitis vinifera и Vitis pseudoreticulata кодируют полипептиды MLO, которые придают восприимчивость грибковому заболеванию мучнистой росе, вызываемой грибом аскомицетом Erysiphe necator. E. necator является облигатным биотрофным грибковым патогеном, которому для размножения требуется растение-хозяин, включая непосредственное взаимодействие между грибом и клетками-хозяевами, чтобы гриб мог получать питательные вещества из растения. Существует множество близко родственных белков MLO, кодируемых семейством генов, каждый из которых уникален для растений и кодирует белки с семью трансмембранными доменами и неизвестной биохимической активностью, локализованные в плазматической мембране. Примечательно, что только специфические гены MLO в семействе способны действовать в качестве генов восприимчивости к мучнистой росе, при этом они кодируют полипептиды с консервативными мотивами в цитоплазматическом C-концевом домене белков MLO. Механизм, посредством которого полипептиды MLO действуют как факторы восприимчивости к мучнистой росе, неизвестен.
Конструкции кпРНК, направленно воздействующие на три разных, но родственных гена MLO видов Vitis, а именно VvMLO6, VvMLO11 и VvMLO15, были сконструированы и получены следующим образом. Например, для первой конструкции была выбрана последовательность гена-мишени VvMLO6 длиной 600 нуклеотидов, соответствующая нуклеотидам 556-1155 SEQ ID NO:138. Были получены химерные ДНК, кодирующие три конструкции кпРНК, направленно воздействующие на гены VvMLO6, VvMLO7 и VvMLO11. Генетические конструкции были созданы с использованием принципов конструирования кпРНК, описанных выше, при этом раздельная последовательность является антисмысловой последовательностью, а сплошная последовательность является смысловой последовательностью (Фигура 1А). Область дцРНК каждой кпРНК имела длину 600 пн; смысловая последовательность в области дцРНК являлась непрерывной сплошной последовательностью, например, соответствовала нуклеотидам 556-1155 SEQ ID NO:138. Нуклеотидная последовательность, кодирующая одну из кпРНК, представлена в настоящем документе в SEQ ID NO:139.
кпРНК получали путем транскрипции in vitro и наносили, отдельно или в виде смеси всех трех, на листья Vitis. Затем споры гриба мучнистой росы наносили на обработанные и необработанные зоны листьев. Даунрегуляцию мРНК-мишени наблюдали с помощью кПЦР и наблюдали развитие заболевания в динамике.
Пример 18. Конструкции РНК, направленно воздействующие на другие гены в растениях
кпРНК, направленно воздействующие на гены TOR A. thaliana и N. benthamiana
Ген Мишень Рапамицина (TOR) кодирует полипептид серин-треониновой протеинкиназы, которая регулирует множество клеточных функций в эукариотических клетках, например в ответ на различные гормоны, стресс и доступность питательных веществ. Он известен как основной регулятор, регулирующий трансляционный аппарат для оптимизации клеточных ресурсов для роста (Abraham, 2002). По меньшей мере, у животных и дрожжей полипептид TOR инактивируется противогрибковым средством рапамицином, что объясняет его обозначение как Мишень Рапамицина (TOR, от англ. Target of Rapamycin). В растениях TOR важен для эмбрионального развития в развивающемся семени, что продемонстрировано летальностью гомозиготных мутантов в TOR (Mahfouz et al., 2006), а также участвует в захвате факторов роста в клеточном метаболизме. Даунрегуляция экспрессии гена TOR, как считают, приводит к увеличению синтеза жирных кислот, приводящему к повышению содержанию липидов в тканях растений.
Конструкции кпРНК, направленно воздействующие на ген TOR Nicotiana benthamiana, нуклеотидная последовательность области кДНК, кодирующей белок, представлена в SEQ ID NO:105, конструировали и получали с использованием принципов конструирования для кпРНК, где раздельная последовательность является смысловой последовательностью, а сплошная последовательность является антисмысловой последовательностью (Фигура 1B). Длина области-мишени составляла 603 нуклеотида, что соответствует нуклеотидам 2595-3197 SEQ ID NO:105. Область дцРНК кпРНК имела длину 603 пн; антисмысловая последовательность в области дцРНК являлась непрерывной сплошной последовательностью, соответствующей комплементу нуклеотидов 2595-3197 SEQ ID NO:105. Нуклеотидные последовательности, кодирующие кпРНК, представлены в настоящем документе в SEQ ID NO:106. Препараты ДНК генетических конструкций, кодирующих конструкции кпРНК, расщепляли рестриктазой MlyI, которая создавала разрыв ДНК сразу после кодирующей области, транскрибировали in vitro с помощью РНК-полимеразы SP6 и очищали РНК, а затем концентрировали в водном буферном растворе. Образцы кпРНК наносили на верхнюю поверхность листьев N. benthamiana. Через 2 дня и 4 дня обработанные образцы листьев собирали, сушили и измеряли общее содержание жирных кислот с помощью количественной газовой хроматографии (ГХ). Образцы листьев, обработанные TOR кпРНК, показали увеличение общего содержания жирных кислот (TFA) с 2,5-3,0% (масса TFA/сухой вес), наблюдаемого в контрольных (необработанных) образцах, до 3,5-4,0% для обработанных кпРНК образцов. Это соответствует увеличению содержания TFA на 17-60% по сравнению с контролем, что указывает на снижение экспрессии гена TOR в тканях, обработанных кпРНК.
кпРНК, направленно воздействующая на ген ALS H. vulgare
Гены ацетолактатсинтазы (ALS) кодируют фермент (EC 2.2.1.6), обнаруженный в растениях и микроорганизмах, катализирующих первую стадию синтеза разветвленных аминокислот лейцина, валина и изолейцина. Фермент ALS катализирует превращение пирувата в ацетолактат, который затем превращается в аминокислоты с разветвленными цепями под действием других ферментов. Ингибиторы ALS используются в качестве гербицидов, таких как гербициды классов сульфонилмочевины, имидазолинона, триазолопиримидина, пиримидинилоксибензоата и сульфониламино-карбонилтриазолинонов.
Для проверки, может ли кпРНК снижать экспрессию гена ALS при экзогенной доставке РНК в растения, была разработана и получена генетическая конструкция, кодирующая кпРНК, которая наравленно воздействовала на ген ALS в ячмене, Hordeum vulgare. Последовательность гена ALS H. vulgare представлена в настоящем документе в SEQ ID NO:107 (рег. номер LT601589). Генетическая конструкция была получена с использованием принципов конструирования кпРНК, при этом раздельная последовательность является смысловой последовательностью, а сплошная последовательность является антисмысловой последовательностью (Фигура 1B). Длина области-мишени составляла 606 нуклеотидов, что соответствует нуклеотидам 1333-1938 SEQ ID NO:107. Область дцРНК кпРНК имела длину 606 пн; антисмысловая последовательность в области дцРНК являлась непрерывной сплошной последовательностью, соответствующей комплементу нуклеотидов 1333-1938 SEQ ID NO:107. Нуклеотидные последовательности, кодирующие кпРНК, представлены в настоящем документе в SEQ ID NO:108. Кодирующая область находилась под контролем промотора РНК-полимеразы SP6 для транскрипции in vitro.
Генетическую конструкцию, кодирующую кпРНК, расщепляли рестриктазой MlyI, которая создавала разрыв после кодирующей области кпРНК, и транскрибировали in vitro с помощью РНК-полимеразы SP6 в соответствии с инструкциями в наборе для транскрипции. РНК наносили на верхнюю поверхность листьев растений ячменя. РНК выделяли из обработанных образцов листьев (через 24 часа). Количественные анализы методом ПЦР с обратной транскрипцией (кПЦР) проводили на образцах РНК. Анализы показали, что уровень мРНК ALS снижался в тканях, обработанных кпРНК. Суммарную РНК выделяли из обработанных и необработанных растений, обрабатывали ДНКазой, проводили количественный анализ и 2 мкг подвергали обратной транскрипции с использованием праймера CTTGCCAATCTCAGCTGGATC. кДНК использовали в качестве матрицы для количественной ПЦР с использованием прямого праймера TAAGGCTGACCTGTTGCTTGC и обратного праймера CTTGCCAATCTCAGCTGGATC. Экспрессию мРНК ALS нормализовали по гену ликопинциклазы Hordeum chilense изолята H1. Экспрессия ALS была снижена на 82% в обработанных LED растениях.
кпРНК, направленно воздействующие на гены NCED1 и NCED2 пшеницы и ячменя
В растениях растительный гормон абсцизовая кислота (АБК) синтезируется из предшественников каротиноидов, причем первая стадия, проходящая в пути синтеза, катализируется ферментом 9-цис-эпокси-каротиноид-диоксигеназой (NCED), которая расщепляет 9-цис-ксантофиллы до ксантоксина (Schwartz et al., 1997). Известно, что гормон АБК способствует состоянию покоя семян (Millar et al., 2006), а также участвует в других процессах, таких как реакция на стресс. Считалось, что повышенная экспрессия гена NCED повышает концентрацию АБК и, таким образом, способствует состоянию покоя. В злаках, таких как пшеница и ячмень, присутствуют два изофермента NCED, обозначенных NCED1 и NCED2, которые кодируют отдельные гомологичные гены.
Для расщепления АБК, фермент АБК-8-гидроксилаза (ABA8OH-2, также известная как CYP707A2), гидроксилирует АБК в качестве стадии в ее катаболизме, что приводит к снятию состояния покоя и протастанию семени.
Конструкции кпРНК, направленно воздействующие на гены, кодирующие HvNCED1 (рег. номер AK361999, SEQ ID NO:109) или HvNCED2 (рег. номер AB239298; SEQ ID NO:110) в ячмене Hordeum vulgare, и соответствующие гомологичные гены в пшенице, были разработаны для трансгенной экспрессии в растениях ячменя и пшеницы. Эти конструкции включали высококонсервативную область генов NCED1 и NCED2 пшеницы и ячменя, причем нуклеотидные последовательности пшеницы и ячменя были приблизительно на 97% идентичны в консервативной области. Генетические конструкции создавали с использованием принципов конструирования кпРНК, описанных выше, причем разделенная последовательность является антисмысловой последовательностью, а сплошная последовательность является смысловой последовательностью (Фигура 1А). Длина области-мишени составляла 602 нуклеотида, что соответствует нуклеотидам 435-1035 последовательности SEQ ID NO:109. Область дцРНК кпРНК имела длину 602 пн; смысловая последовательность в области дцРНК являлась непрерывной сплошной последовательностью, соответствующей нуклеотидам 435-1035 SEQ ID NO:110. Нуклеотидные последовательности, кодирующие кпРНК NCED1 и NCED2, представлены в настоящем документе в SEQ ID NO:111 и 112.
Аналогичным образом, была получена конструкция кпРНК, направленно воздействующая на ген ABA-OH-2 пшеницы T. aestivum и ячменя H. vulgare (рег. номер DQ145933, SEQ ID NO:113). Область-мишень имела длину 600 нуклеотидов, соответствующих нуклеотидам 639-1238 SEQ ID NO:113. Область дцРНК кпРНК имела длину 600 пн; смысловая последовательность в области дцРНК была непрерывной, сплошной последовательностью, соответствующей нуклеотидам 639-1238 SEQ ID NO:113. Нуклеотидная последовательность химерной ДНК, кодирующей кпРНК, представлена в SEQ ID NO:114.
Химерные ДНК, кодирующие кпРНК, встраивали в вектор экспрессии под контролем промотора гена Ubi, конститутивно экспрессирующегося в большинстве тканей, в том числе в развивающемся семени. Кассеты экспрессии вырезали и встраивали в бинарный вектор. Их использовали для получения трансформированных растений пшеницы.
Трансгенные растения пшеницы выращивали до зрелости, из них получали семена и исследовали на пониженную экспрессию генов NCED или ABA-OH-2 и влияние на состояние покоя зерна, соответствующее сниженной экспрессии генов. Ожидали ряд фенотипов по степени изменения состояния покоя. Для модуляции степени измененных фенотипов создавали модифицированные генетические конструкции для экспрессии кпРНК, содержащих пары оснований G:U в областях двухцепочечной РНК, особенно для кпРНК, где примерно 15-25% нуклеотидов в области двухцепочечных кпРНК участвуют в спаривании оснований G:U в процентах от общего числа нуклеотидов в двухцепочечной области.
кпРНК, направленно воздействующие на ген EIN2 A. thaliana
Как описано в Примере 10, ген EIN2 Arabidopsis thaliana кодирует рецепторный белок, участвующий в реагировании на этилен. Рассада мутанта EIN2 демонстрирует удлинение гипокотиля по сравнению с рассадой дикого типа при проращивании на ACC. Так как ген экспрессируется в ростках вскоре после прорастания семян, доставку кпРНК трансгенными средствами считали наиболее подходящим методом для проверки степени даунрегуляции EIN2 в сравнении с экзогенной доставкой предварительно полученной РНК.
Была разработана конструкция кпРНК, направленно воздействующая на ген EIN2 Arabidopsis thaliana (SEQ ID NO:115) в области длиной 400 нуклеотидов мРНК гена-мишени. Конструкцию получали путем вставки последовательности (SEQ ID NO:116), кодирующей кпРНК, в вектор, включающий 35S промотор для экспрессии кпРНК в растениях A. thaliana. Трансгенные растения A. thaliana получали и тестировали на снижение экспрессии гена EIN2 с помощью кПЦР и анализа длины гипокотиля в присутствии ACC. Снижение уровней экспрессии EIN2 и увеличение длины гипокотиля наблюдается в растениях некоторых трансгенных линий.
кпРНК, направленно воздействующие на ген CHS A. thaliana
Ген халконсинтазы (CHS) в растениях кодирует фермент, катализирующий превращение 4-кумароил-КоА и малонил-КоА в нарингенин-халкон, который является первым ферментом, участвующим в биосинтезе флавоноидов. Флавоноиды являются классом органических соединений, обнаруженных в основном в растениях, участвующих в защитных механизмах и устойчивости к стрессам.
Была разработана конструкция кпРНК, направленно воздействующая на ген CHS Arabidopsis thaliana (SEQ ID NO:117) в области из 338 нуклеотидов мРНК ген-мишени. Конструкцию получали путем вставки последовательности ДНК (SEQ ID NO:118), кодирующей кпРНК, в вектор, включающий 35S промотор для экспрессии кпРНК в растениях A. thaliana. Трансгенные растения A. thaliana получали путем трансформации генетической конструкцией в бинарном векторе и проверяли на снижение экспрессии гена CHS с помощью кПЦР и на снижение продукции флавоноидов. Снижение уровней экспрессии CHS и снижение уровней флавоноидов наблюдается в растениях некоторых трансгенных линий, например, в семенной оболочке трансгенных семян.
кпРНК, направленно воздействующая на ген LanR Lupinus angustifolius
Ген LanR узколистного люпина, Lupinus angustifolius L., кодирует полипептид, который является родственным по последовательности N-гену табака, который придает устойчивость к вирусному заболеванию, вызываемому вирусом табачной мозаики (ВТМ).
Была сконструирована и получена химерная ДНК для продукции молекулы кпРНК, направленно воздействующей на ген LanR L. angustifolius (рег. номер XM_019604347, SEQ ID NO:119). Генетическая конструкция была получена с помощью принципов конструирования кпРНК, описанных выше, при этом разделенная последовательность являлась антисмысловой последовательностью, а сплошная последовательность являлась смысловой последовательностью (Фигура 1A). Нуклеотидная последовательность, кодирующая кпРНК, представлена в настоящем документе в SEQ ID NO:120. кпРНК получали путем транскрипции in vitro, очищали и концентрировали, и аликвоты РНК наносили на листья растений L. angustifolius, содержащих ген LanR. Образцы вируса наносили на обработанные и необработанные растения и через несколько дней сравнивали симптомы заболевания.
Пример 19. Конструкции РНК, направленно воздействующие на ген насекомого
Введение
Тли являются насекомыми, которые питаются соком растений и наносят существенный, и иногда серьезный ущерб растениям непосредственно в результате питания растительным соком, а в некоторых случаях - косвенно, путем передачи различных вирусов, вызывающих болезни у растений. Хотя Bt-токсин в некоторых случаях был эффективен для защиты сельскохозяйственных растений от грызущих насекомых, он, как правило, не был эффективен против насекомых, питающихся соком растений. Сорта растений, содержащие гены устойчивости, могут быть эффективным способом борьбы с тлей, однако большинство генов устойчивости являются высокоспецифичными к определенным видам или биотипам тли, и устойчивость часто преодолевается вследствие быстрой эволюции новых биотипов посредством генетических или эпигенетических изменений. Более того, гены устойчивости недоступны во многих культурных растениях или могут отсутствовать для некоторых видов многоядных тлей, таких как зеленая персиковая тля, которые поражают широкий спектр хозяев. В настоящее время борьбу с тлей осуществляют главным образом путем частого применения пестицидов, что приводит к устойчивости тли к пестицидам. Например, в Австралии против зеленой персиковой тли эффективной осталась только одна группа пестицидов по механизму действия, поскольку ей удалось приобрести устойчивость ко всем остальным зарегистрированным инсектицидам.
В нескольких исследованиях было показано, что РНКи-опосредованный сайленсинг генов может быть полезен в качестве инструмента исследования в ряде видов тли, см. обзоры Scott et al., 2013; Yu et al., 2016, но не было показано, что он эффективно защищает растения от тли. В этих исследованиях дцРНК, направленно воздействующие на ключевые гены, участвующие в росте и развитии тли, процессах заражения или питания, доставляли путем прямой инъекции тлям или путем питания тлей искусственными диетами, содержащими дцРНК.
Для проверки потенциала модифицированных молекул РНКи, таких как молекулы кпРНК, описанные в настоящем документе, в борьбе с насекомыми, питающимися соком растений, авторы изобретения выбрали зеленую персиковую тлю (Myzus persicae) в качестве модельного сокососущего насекомого по нескольким причинам. Во-первых, зеленая персиковая тля - это многоядное насекомое, которое поражает широкий спектр видов растений-хозяев, включая основные зерновые и плодоовощные культуры по всему миру. Во-вторых, зеленая персиковая тля является переносчиком некоторых опасных вирусов, таких как вирус западной желтухи свеклы, который наносит большой ущерб в некоторых районах выращивания канолы. Для этого исследования в качестве генов-мишеней для даунрегуляции были выбраны два гена тли, один из которых кодирует ключевой эффекторный белок (C002), а второй кодирует рецептор активированной протеинкиназы C (Rack-1). Белок C002 представляет собой белок слюнной железы тли, который необходим для питания тли на своем растении-хозяине (Mutti et al., 2006; Mutti et al., 2008). Rack1 является внутриклеточным рецептором, который связывает активированную протеинкиназу C, фермент, который в основном участвует в каскадах сигнальной трансдукции (McCahill et al., 2002; Seddas et al., 2004). MpC002 преимущественно экспрессируется в слюнной железе тли, а MpRack1 преимущественно экспрессируется в кишечнике. В предыдущих исследованиях применение РНКи путем прямой инъекции или кормления искусственной диетой привело к гибели нескольких тестируемых видов тлей (Pitino et al., 2011; Pitino and Hogenhout, 2012; Yu et al., 2016).
Материалы и методы: Культура тлей и растительные материалы
Зеленых персиковых тлей (Myzus persicae) собирали в Западной Австралии. Перед каждым экспериментом тлей разводили на растениях редьки (Raphanus sativus L.) при естественном освещении в комнате инсектария. Тлей переносили в экспериментальные клетки с искусственной диетой с помощью тонкой кисти.
Компоненты искусственной диеты для кормления тлей были такими же, как описано в публикации Dadd and Mittler (1966). В устройстве для искусственного кормления тлей использовалась пластиковая трубка диаметром 1 см и высотой 1 см. Искусственную диету для тлей, 100 мкл, с или без кпРНК, помещали между двумя слоями парафильма для создания пищевого пакетика. Сверху на этом пакете помещали камеру для тлей, которые могли перемещаться и питаться диетой, протыкая своими хоботками верхний слой натянутого парафильма. Восемь нимф первого или второго возраста осторожно переносили в камеру для тлей с помощью тонкой кисти. Эксперимент проводили в климакамере при 20°C.
Листья табака, используемые в одном эксперименте, собирали с растений, выращенных в почве в условиях цикла 16 ч света/8 ч темноты при 22°C.
Гены MpC002 и MpRack-1 и конструкции кпРНК
Гены зеленой персиковой тли MpC002 и MpRack-1, тестируемые в качестве генов-мишеней, были такими же, как описано в публикации Pitino et al. (2011; 2012). Последовательности ДНК обоих генов были получены с веб-сайта NCBI, MpC002 (>MYZPE13164_0_v1.0_000024990.1 | 894 нт) и MpRack-1 (>MYZPE13164_0_v1.0_000198310.1 | 960 нт). Последовательности кДНК двух генов представлены в настоящем документе в SEQ ID NO:123 и 124. Конструкции кпРНК были сконструированы так же образом, как описано в предыдущих примерах. Последовательности ДНК, кодирующие молекулы кпРНК, представленные в настоящем документе в SEQ ID NO:125 и 126, использовали в качестве матриц транскрипции для синтеза кпРНК. Векторные ДНК, кодирующие молекулы кпРНК, направленно воздействующие на гены MpC002 и MpRack-1, вводили в штамм E. coli DH5α для получения плазмидной ДНК для транскрипции РНК in vitro и в штамм Е.coli HT115 для транскрипции in vivo (в бактериях).
Эффективность молекул кпРНК при снижении продуктивности тли
Для исследования, влияют ли кпРНК, направленно воздействующие на гены MpC002 или MpRack-1, на продуктивность тли, каждую кпРНК доставляли тлям посредством искусственной диеты, как описано в Примере 1. В каждый эксперимент включали десять биологических повторностей; каждая биологическая повторность включала по восемь нимф зеленой персиковой тли первой - второй возрастной стадии. В качестве контроля в каждом эксперименте использовали эквивалентные концентрации нерелевантной кпРНК, а именно ledGFP.
При более низкой концентрации 50 нг/мкл каждой молекулы кпРНК выживаемость тли после кормления искусственной диетой, содержащей кпРНК MpC002 или MpRack-1, существенно не отличалась от контрольной ledGFP. Однако кпРНК, направленно воздействующая на ген MpC002, значимо (P<0,05) снижала скорость размножения зеленой персиковой тли (Фигура 37А). Среднее число нимф от взрослой тли было уменьшено примерно на 75% по сравнению с количеством нимф у имаго, которых содержали на контрольной диете с контрольной кпРНК. При более высокой концентрации, 200 нг/мкл, кпРНК, направленно воздействующие либо на MpC002, либо на MpRack-1, увеличивали летальность у взрослых тлей (Фигура 37B). Снижение выживаемости тли на диетах, включающих кпРНК MpC002 или MpRack-1, также наблюдали через 24 часа и в течение пятидневного периода эксперимента. Результаты показали, что применение кпРНК, направленно воздействующих на жизненно важные гены тли, могло вызывать гибель тли и уменьшать размножение тли. Эффективность каждой кпРНК сравнивали с молекулой шпилечной РНК (шРНКи), направленной на ту же область гена-мишени.
Захват молекул кпРНК тлями
Для отслеживания захвата и распределения молекул кпРНК в организме тлей, кпРНК, направленно воздействующие на гены MpC002 или MpRack-1, метили Cy3 (меченными цианиновым красителем нуклеотидтрифосфатами) в процессе синтеза, как описано в примере 1. Мечение Cy3, согласно имеющимся сообщениям, не влияет на биологическую функцию обычных молекул дцРНК и поэтому может использоваться в качестве метки для детектирования по флуоресценции. Тлей, которые получали с питанием меченые кпРНК, исследовали с помощью конфокальной микроскопии при использовании микроскопа Leica EL6000. Меченную Cy3 кпРНК, направленно воздействующую на MpC002 или MpRack-1, обнаруживали в кишечнике тли в течение нескольких часов после кормления на искусственной диете, а затем в репродуктивной системе и даже у новорожденных нимф, которые являлись потомством зрелых особей, получавших диету. Результаты показали, что гены тли, критически важные для функции пищеварительной системы или репродукции, могут быть эффективными мишенями для молекул кпРНК посредством питания.
Стабильность кпРНК
Для исследования стабильности кпРНК в диете и после выделения из получавших диету тлей, РНК выделяли из искусственной диеты и из медвяной пади тлей после питания диетами, содержащими меченые молекулы кпРНК. Образцы РНК подвергали электрофорезу в гелях и исследовали с помощью флуоресцентного детектирования. РНК ledMpC002 до питания четко отображала один продукт примерно 700 пн в агарозном геле. РНК, выделенная из искусственной диеты, показала шмер РНК размером 100-700 пн, что указывает на некоторую деградацию после контакта с диетой при комнатной температуре в течение 25 дней, но все еще в значительной степени интактную. РНК, выделенная из медвяной пади тли, показала флуоресценцию в диапазоне РНК от 350 до 700 пн, поэтому опять же была в значительной степени интактной. Несмотря на деградацию некоторых кпРНК, значительная часть молекул кпРНК была способна оставаться интактной в искусственной диете, а также в медвяной пади тли в течение значительного периода времени. Эта степень стабильности молекул кпРНК должна позволять кпРНК оставаться активной и сохранять активность при экзогенном применении.
Поглощение меченой кпРНК листьями растений
Cy3-меченную РНК ledMpC002 наносили кистью на верхнюю поверхность листьев табака с целью наблюдения, могла ли она проникать через ткани листа. Десять микролитров Cy3-меченной ledMpC002 (концентрация 1 мкг/мл) наносили в круг диаметром 2 см и отмечали область нанесения черным маркером. Снимки флуоресценции листьев при возбуждении 525 нм делали в течение пятичасового периода с помощью микроскопа Leica EL6000, сравнивая окрашеные ткани с неокрашеными. Метка Cy3 поддавалась четкому обнаружению в паренхиме листа в течение одного часа после применения, то есть явно проникала через восковой слой кутикулы на поверхности листа. Уровень флуоресценции увеличился через 2 часа и сохранялся до 5 часов. Было неясно, проникли ли молекулы кпРНК в клетки или в ядра клеток. Однако, поскольку сокососущие насекомые питаются исключительно из ситовидных элементов флоэмы листьев и стеблей растения, перенос РНК в растительные клетки не требовался для сайленсинга генов тли. Эксперимент показал, что молекулы кпРНК были обнаружены в растительных тканях после нанесения на поверхность.
Выводы
Цель данного исследования состояла в том, чтобы протестировать применение экзогенной РНКи с использованием конструкции кпРНК для борьбы с тлями, основной группой сокососущих насекомых-вредителей, которые являются проблемой во всем мире. Известно, что тля обладает аппаратом РНКи для процессинга экзогенной РНК (Scott et al., 2013; Yu et al., 2016). В данном случае пероральная доставка через искусственную диету, содержащую молекулы кпРНК, направленные на гены MpC002 или MpRack-1, была способна вызвать гибель тли и уменьшить размножение тли. Молекулы были протестированы против двух разных генов-мишеней, один из которых кодирует эффекторный белок C002, а другой - рецептор активированной протеинкиназы (Rack-1), которые необходимы для питания и развития зеленой персиковой тли (Myzus persicae). При добавлении в искусственную диету в концентрации до 50 нг/мкл, молекулы кпРНК, направленно воздействующие на эти гены, значительно снижают размножение тли. При более высокой концентрации, составляющей 200 нг/мкл, кпРНК также увеличивали гибель тли. При исследовании поглощения кпРНК с использованием мечения Cy3, молекулы кпРНК наблюдали в кишечнике тли в течение нескольких часов после поглощения искусственной диеты, а затем в репродуктивной системе и даже в новорожденных нимфах, которые были потомством зрелых особей, получавших диету.
Также было показано, что молекулы кпРНК оставались в значительной степени интактными в искусственной диете в течение по меньшей мере трех с половиной недель. По существу интактные молекулы кпРНК также были обнаружены в медвяной росе тли, продукте, выделяемом тлей. При нанесении меченой кпРНК на листья растений, она могла попадать во флоэму, из которой питаются тли. В совокупности эти результаты указывают на высокий потенциал применения кпРНК для борьбы с тлей и другими сокососущими насекомыми, в том числе путем экзогенной доставки через пищу, обеспечивая практический подход к борьбе с тлями и другими сокососущими насекомыми. Эти молекулы РНК также могут экспрессироваться в трансгенных растениях при использовании промоторов, которые способствуют синтезу РНК в тканях флоэмы, для борьбы с тлей и другими сокососущими насекомыми.
Пример 20. Конструкции РНК, направленно воздействующие на другие гены насекомых
кпРНК, направленно воздействующие на гены насекомых-вредителей
Helicoverpa armigera является насекомым-вредителем из отряда Чешуекрылых, также известным как совка хлопковая. Личинки H. armigera питаются широким спектром растений включая многие важные культурные зерновые культуры и приносят значительный ущерб урожаю стоимостью в миллиарды долларов в год. Личинки являются многоядными и космополитными вредителями, которые могут питаться широким спектром растений, включая хлопок, кукурузу, помидор, нут, голубиный горох, люцерну, рис, сорго и коровий горох.
Ген ABC-переносчика white H. armigera (ABCwhite) был выбран в качестве гена-мишени с легко обнаруживаемым фенотипом для проверки конструкций кпРНК и кпРНК(G:U) в личинке насекомого. Белки переносчики ABC (от англ. ATP Binding Cassette - АТФ-связывающая кассета) относятся к суперсемейству переносчиков АТФ-связывающей кассеты, например, в геноме Helicoverpa были идентифицированы 54 различных гена ABC-переносчика. Переносчики ABC кодируют мембраносвязанные белки, которые переносят одну или более из целого ряда молекул через мембраны. Белки используют энергию, высвобождаему при гидролизе АТФ, для транспорта молекул через мембрану. Некоторые ABC-переносчики участвовали в деградации растительных вторичных метаболитов у хлопковой совки, H. armigera (Khan et al., 2017). Белок ABCwhite переносит предшественники пути оммохромов и птеридинов в пигментные гранулы в глазах, поэтому нокаутные мутанты имеют белые глаза.
Нуклеотидная последовательность гена ABCwhite представлена в SEQ ID NO:127 (рег. номер KU754476). Для проверки, может ли кпРНК снижать экспрессию гена ABCwhite при экзогенной доставке РНК в пищу личинок, была разработана и получена генетическая конструкция, кодирующая кпРНК, которая направленно воздействует на ген. Генетическая конструкция была получена с использованием принципов конструирования кпРНК, при этом раздельная последовательность является смысловой последовательностью, а сплошная последовательность является антисмысловой последовательностью (Фигура 1B). Длина области-мишени составляла 603 нуклеотида, что соответствует нуклеотидам 496-1097 SEQ ID NO:127. Область дцРНК кпРНК имела длину 603 пн; антисмысловая последовательность в области дцРНК являлась непрерывной сплошной последовательностью, соответствующей комплементу нуклеотидов 496-1097 SEQ ID NO:127. Нуклеотидные последовательности, кодирующие кпРНК, представлены в настоящем документе в SEQ ID NO:128. Кодирующая область находилась под контролем промотора РНК-полимеразы Т7 для транскрипции in vitro.
Генетическую конструкцию, кодирующую кпРНК, расщепляли эндонуклеазой рестрикции SnaBI, которая создает разрыв после кодирующей кпРНК области, и транскрибировали in vitro с использованием РНК-полимеразы T7 согласно инструкциям в наборе для транскрипции. РНК добавляли в искусственную диету и давали личинкам H. armigera.
Соответствующую конструкцию кпРНК, содержащую пары оснований G:U в двухцепочечном стебле, получали и сравнивали с канонически спаренной кпРНК.
кпРНК, направленно воздействующие на ген у муравьев
Linepithema humile, обычно известный как аргентинский муравей, является насекомым-вредителем, широко распространившимся на нескольких континентах. Ген, кодирующий нейропептид активации биосинтеза феромонов (PBAN) L. humile, подобный нейропептидам (LOC105673224), был выбран в качестве гена-мишени, участвующий в коммуникации между насекомыми посредством феромонов.
Нуклеотидная последовательность гена PBAN представлена в SEQ ID NO:129 (рег. номер XM_012368710). Для проверки, может ли кпРНК снижать экспрессию гена PBAN при экзогенной доставке РНК с пищей в форме приманки, была разработана и получена генетическая конструкция, кодирующая кпРНК, которая направленно воздействует на ген. Генетическая конструкция была получена с использованием принципов конструирования кпРНК, при этом раздельная последовательность является смысловой последовательностью, а сплошная последовательность является антисмысловой последовательностью (Фигура 1B). Длина области-мишени составляла 540 нуклеотидов, что соответствует нуклеотидам 136-675 SEQ ID NO:129. Область дцРНК кпРНК имела длину 540 пн; антисмысловая последовательность в области дцРНК являлась непрерывной сплошной последовательностью, соответствующей комплементу нуклеотидов 136-675 SEQ ID NO:129. Нуклеотидные последовательности, кодирующие кпРНК, представлены в настоящем документе в SEQ ID NO:130. Кодирующая область находилась под контролем промотора РНК-полимеразы Т7 для транскрипции in vitro.
Генетическую конструкцию, кодирующую кпРНК, расщепляли эндонуклеазой рестрикции SnaBI, которая создает разрыв после кодирующей кпРНК области, и транскрибировали in vitro с использованием РНК-полимеразы T7 согласно инструкциям в наборе для транскрипции. РНК наносили на кукурузную муку для пероральной доставки муравьям L. humile.
кпРНК, направленно воздействующие на гены L. cuprina
Lucilia cuprina - насекомое-вредитель, более известное как австралийская зеленая мясная муха. Оно относится к семейству мясных мух, Calliphoridae, и является членом отряда насекомых Diptera. Пять генов-мишеней были выбраны для тестирования с конструкциями кпРНК, а именно гены, кодирующие каталитическую субъединицу A протонной АТФазы V-типа (рег. номер XM_023443547), РНКазу 1/2 (рег. номер XM_023448015), хитинсинтазу (рег. номер XM_023449557), рецептор экдизона (EcR; рег. номер U75355) и гамма-тубулин 1/1-подобный (рег. номер XM_023449717) L. cuprina. Каждая из генетических конструкций была получена с использованием принципов конструирования кпРНК, при этом раздельная последовательность является смысловой последовательностью, а сплошная последовательность является антисмысловой последовательностью (Фигура 1B). В каждом случае область-мишень имела длину около 600 нуклеотидов, а антисмысловая последовательность в области дцРНК являлась непрерывной сплошной последовательностью. Нуклеотидная последовательность, кодирующая кпРНК, направленно воздействующую на ген АТФазы-A, представлена в настоящем документе в SEQ ID NO:131. Нуклеотидная последовательность, кодирующая кпРНК, направленно воздействующую на ген РНКазы 1/2, представлена в настоящем документе в SEQ ID NO:132. Нуклеотидная последовательность, кодирующая кпРНК, направленно воздействующую на ген хитинсинтазы, представлена в настоящем документе в SEQ ID NO:133. Нуклеотидная последовательность, кодирующая кпРНК, направленно воздействующую на ген EcR, представлена в настоящем документе в SEQ ID NO:134. Нуклеотидная последовательность, кодирующая кпРНК, направленно воздействующую на ген, подобный 1/1 гамма-тубулину, представлена в настоящем документе в SEQ ID NO:135. В каждой конструкции кодирующая область находилась под контролем промотора РНК-полимеразы Т7 для транскрипции in vitro.
Для специалистов в данной области будет очевидно, что в изобретение могут быть внесены многочисленные изменения и/или модификации, как показано в конкретных вариантах осуществления, без отступления от сущности или объема изобретения, которые описаны в общем виде. Таким образом, настоящие варианты осуществления следует рассматривать во всех отношениях как иллюстративные, а не ограничивающие.
Настоящая заявка испрашивает приоритет AU 2017903773, поданной 15 сентября 2017 года, AU 2018902840, поданной 3 августа 2018 года, и AU 2018902896, поданной 8 августа 2018 года, описания которых включены в настоящий документ посредством отсылки.
Все публикации, обсуждаемые и/или цитируемые в настоящем документе, включены в настоящий документ во всей своей полноте.
Любые обсуждения документов, действий, материалов, устройств, изделий или т.п., которые включены в настоящее описание, предназначены исключительно для обеспечения контекста для настоящего изобретения. Не следует воспринимать как допущение того, что какие-либо или все эти положения являются частью предшествующего уровня техники или были общеизвестными в области, относящейся к настоящему изобретению, поскольку они существовали до даты приоритета каждого пункта формулы настоящей заявки.
ИСПОЛЬЗОВАННЫЕ ИСТОЧНИКИ
Abraham (2002). Cell 111:9-12.
Acevedo-Garcia et al., (2017). Plant Biotechnology Journal 15:367-378.
Alvarez et al., (2000). Theor Appl Genet 100:319-327.
Baumlein et al., (1991). Mol. Gen. Genet. 225:459-467.
Baumlein et al., (1992). Plant J. 2:233-239.
Bhattacharyya et al., (1990) Cell 60:155-122.
Brar et al., (1996) Biotech Genet. Eng Rev 13:167-79.
Broothaerts et al., (2005). Nature 433:629-633.
Broun et al., (1998). Plant J. 13:201-210.
Busk et al., (1997). Plant J. 11:1285-1295.
Chen et al., (2005). Functional Plant Biology 32:671-681.
Chikwamba et al., (2003). Proc. Natl. Acad. Sci. U.S.A. 100:11127-11132.
Christou and Klee, Handbook of Plant Biotechnology, John Wiley and Sons (2004).
Chung et al., (2006). BMC Genomics 7:120.
Clough and Bent (1998). Plant J. 16:735-743.
Corrado and Karali (2009). Biotechnol. Adv. 27:733-743.
Courvalin et al., (1995). Life Sci. 318:1209-1212.
Dadd and Mittler (1966). Experientia 22:832-833.
Darji et al., (1997). Cell 91:765-775.
Dong et al., (2011) Plant J. 68:633-45.
Draper and Scott (1988). In: J. Draper et al., (Eds.), Plant Genetic Transformation and Gene Expression: A Laboratory Manual, Alden Press, Oxford, pp. 199-236.
Dunwell (2000). J Exp Botany 51Spec No:487-496.
Ellerstrom et al., (1996). Plant Mol. Biol. 32:1019-1027.
Elliott et al., (2002). Mol. Plant Microbe Interact. 15:1069-1077.
Ellis et al., (1987). EMBO J 6:11-16.
Fehr, In: Breeding Methods for Cultivar Development, Wilcox J. ed., American Society of Agronomy, Madison Wis. (1987).
Gan (1995). Molecular characterization and genetic manipulation of plant senescence. PhD thesis. University of Wisconsin, Madison.
Gan and Amasino (1995). Science 270:1986-1988.
Glawe (2008). Ann. Rev. Phytopathol. 46:27-51.
Gleave (1992). Plant Mol Biol 20: 1203-1207.
Gupta et al., (1988) Plant Mol. Biol. 10:215-224.
Helliwell and Waterhouse (2005). Methods in Enzymology 392:24-35.
Hinchee et al., (1988). Biotechnology 6:915-922.
Horvath et al., (2000). Proc. Natl. Acad. Sci. U.S.A. 97:1914-1919.
Hsieh and Fire (2000) Annu Rev Genet 14:187-204.
Jeddeloh, et al., (1999). Nat. Genet. 22:94-97.
Jefferson et al., (1987). EMBO J 6:3901-3907.
Jepson et al., (1994). Plant Mol. Biol. 26:1855-1866.
Khan (2017). Sci Rep 7, 40025
Kishore and Somerville (1993). Curr Opin Biotechnol. 4:152-158.
Koziel et al., (1996). Plant Mol. Biol. 32:393-405.
Lacroix et al., (2008). Proc. Natl. Acad. Sci.U.S.A. 105: 15429-15434.
Li et al., (1996). FEBS Lett. 379:117-121.
McCahill et al., (2002). Molecular Pharmacology 62:1261-1273.
McCullough and Schuler (1997). Nucl Acids Res. 25:1071-1077.
Mahfouz et al., (2006). Plant Cell 18:477-490.
Matsuoka et al., (1994). Plant J. 6:311-319.
Meier et al., (1997). FEBS Lett. 415:91-95.
Melamed-Bessudo et al., (2012) Proc. Natl. Acad. Sci. USA 109(16):e981-988.
Millar et al., (2006). Plant J. 45:942-954.
Mutti et al., (2006). J Insect Sci 6:38.
Mutti et al., (2008). Proceedings of the National Academy of Sciences 105:9965-9969.
Olive et al., (1989) Plant Mol Biol 12:525-538.
Padidam (2003). Transgenic Res. 12:101-109.
Perrin et al., (2000). Mol Breed 6:345-352.
Pitino et al., (2011). PLoS ONE 6, e25709.
Pitino and Hogenhout (2012). Molecular Plant-Microbe Interactions 26:130-139.
Potenza et al., (2004). In Vitro Cell Dev. Biol. Plant 40:1-22.
Powell et al., (1996). Vaccines 183, Abstract.
Preiss et al., (1987). In: Tailoring Genes for Crop Improvement (Bruening et al., eds.), Plenum Press, S.133-152.
Sambrook et al., (1989). Molecular Cloning: A Laboratory Manual, Cold Spring Harbour Laboratory Press.
Schaffner (1980). Proc. Natl. Acad. Sci. U.S.A. 77:2163-2167.
Schwartz et al., (1997). Science 276:1872-1874.
Scott et al., (2013). Journal of Insect Physiology 59:1212-1221.
Seddas et al., (2004). Virology 325:399-412.
Shiina et al., (1997). Plant Physiol. 115:477-483.
Shure et al., (1983). Cell 35:225-233.
Sizemore et al., (1995). Science 270:299-302.
Slater et al., Plant Biotechnology - The Genetic Manipulation of Plants, Oxford University Press (2003).
Smith et al., (2000). Nature 407:319-320.
Stalker et al., (1988). Science 242: 419-423.
Stewart et al., (2000). J Mol Biol 298:611-622.
Tan et al., (2011). Plant Physiol. 156:1577-1588.
Thillet et al., (1988). J. Biol. Chem 263:12500-12508.
Timmons et al., (2001). Gen 263:103-112.
Ulmasov et al., (1995). Plant Physiol. 108:919-927.
Wang MB. Isolation of phloem specific gene promoters for use in genetic engineering of insect resistance in rice. PhD thesis, University of Durham, UK.
Wang et al., (1994) Plant Molecular Biology 24:159-170.
Wang et al., (1998) Acta Horticulturae 461:401-407.
Wang et al., (2008) RNA 14: 903-913.Wang et al., (2013). PLoS Genet 9, e1003865.
Wang et al., (2014). Nature Biotechnology 32:9.
Weiss (2003). Int. J. Med. Microbiol. 293:95:106.
Weissbach et al., (1988). In: Methods for Plant Molecular Biology, Academic Press, San Diego, Calif.
Wijnker et al., (2008). Trends in Plant Science 13:640-646.
Yang et al., Particle Bombardment Technology for Gene Transfer, Oxford Press, Oxford, England (1994).
Yang et al., (2003). Planta 216:597-603.
Yelina et al., (2012). PLoS Genetics 8(8):e1002844. doi:10.1371/journal.pgen.1002844
Yu et al., (2016). Pest Management Science 72:1090-1098.
Zhang et al., (2018). Nat Rev Mol Cell Biol. 19:489-506.
--->
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> КОММОНВЕЛТ САЙЕНТИФИК ЭНД ИНДАСТРИАЛ РИСЕРЧ ОРГАНИЗЕЙШН
(COMMONWEALTH SCIENTIFIC AND INDUSTRIAL RESEARCH ORGANISATION)
<120> Молекулы РНК
<130> 525258PCT
<150> 2018902896
<151> 2018-08-08
<150> 2018902840
<151> 2018-08-03
<150> 2017903773
<151> 2017-09-15
<160> 139
<170> PatentIn version 3.5
<210> 1
<211> 1229
<212> РНК
<213> Искусственная последовательность
<220>
<223> GFP кпРНК
<400> 1
gggugucgcc cucgaacuuc accucggcgc gggucuugua guugccgucg uccuugaaga
60
agauggugcg cuccuggacg uagccuucgg gcauggcgga cuugaagaag ucgugcugcu
120
ucaugugguc gggguagcgg cugaagcacu gcacgccgua ggugaaggug gucacgaggg
180
ugggccaggg cacgggcagc uugccggugg ugcagaugaa cuucaggguc agcuugccgu
240
agguggcauc gcccucgccc ucgccggaca cgcugaacuu guggccguuu acgucgccgu
300
ccagcucgac caggaugggc accaccccgg ugaacagcuc cucgcccuug cucacuaugg
360
aucaacuagg gaucccccug aaguucaucu gcaccaccgg caagcugccc gugcccuggc
420
ccacccucgu gaccaccuuc accuacggcg ugcagugcuu cagccgcuac cccgaccaca
480
ugaagcagca cgacuucuuc aaguccgcca ugcccgaagg cuacguccag gagcgcacca
540
ucuucuucaa ggacgacggc aacuacaaga cccgcgccga ggugaaguuc gagggcgaca
600
cccuggugaa ccgcaucgag cugaagggca ucgacuucaa ggaggacggc aacauccugg
660
ggcacaagcu ggaguacaac uacaacagcc acaacgucua uaucauggcc gacaagcaga
720
agaacggcau caaggugaac uucaagaucc gccacaacau cgaggacggc agcgugcagc
780
ucgccgacca cuaccagcag aacaccccca ucggcgacgg ccccgugcug cugccaagcu
840
uuaggugauc caagcuugau ccgggcuuua cuuguacagc ucguccaugc cgagagugau
900
cccggcggcg gucacgaacu ccagcaggac caugugaucg cgcuucucgu uggggucuuu
960
gcucagggcg gacugggugc ucagguagug guugucgggc agcagcacgg ggccgucgcc
1020
gaugggggug uucugcuggu aguggucggc gagcugcacg cugccguccu cgauguugug
1080
gcggaucuug aaguucaccu ugaugccguu cuucugcuug ucggccauga uauagacguu
1140
guggcuguug uaguuguacu ccagcuugug ccccaggaug uugccguccu ccuugaaguc
1200
gaugcccuuc agcucgaugc gguucacca
1229
<210> 2
<211> 1326
<212> РНК
<213> Искусственная последовательность
<220>
<223> GUS кпРНК
<400> 2
gggaacagac gcgugguuac agucuugcgc gacaugcguc accacgguga uaucguccac
60
ccagguguuc ggcguggugu agagcauuac gcugcgaugg auuccggcau aguuaaagaa
120
aucauggaag uaagacugcu uuuucuugcc guuuucgucg guaaucacca uucccggcgg
180
gauagucugc caguucaguu cguuguucac acaaacggug auacguacac uuuucccggc
240
aauaacauac ggcgugacau cggcuucaaa uggcguauag ccgcccugau gcuccaucac
300
uuccugauua uugacccaca cuuugccgua augagugacc gcaucgaaac gcagcacgau
360
acgcuggccu gcccaaccuu ucgguauaaa gacuucgcgc ugauaccaga cgugccguau
420
guuauugccg ggaaaagugu acguaucacc guuuguguga acaacgaacu gaacuggcag
480
acuaucccgc cgggaauggu gauuaccgac gaaaacggca agaaaaagca gucuuacuuc
540
caugauuucu uuaacuaugc cggaauccau cgcagcguaa ugcucuacac cacgccgaac
600
accugggugg acgauaucac cguggugacg caugucgcgc aagacuguaa ccacgcgucu
660
guucccgacu ggcagguggu ggccaauggu gaugucagcg uugaacugcg ugaugcggau
720
caacaggugg uugcaacugg acaaggcacu agcgggacuu ugcaaguggu gaauccgcac
780
cucuggcaac cgggugaagg uuaucucuau gaacugugcg ucacagccaa aagccagaca
840
gagugugaua ucuacccgcu ucgcgucggc auccggucag uggcagugaa gggccaacag
900
uuccugauua accacaaacc guucuacuuu acuggcuuug gucgucauga agaugcggac
960
uuacguggca aaggauucga uaacgugcug auggugcacg accacgcauu aauggacugg
1020
auuggggcca acuccuaccg uaccucgcau uacccuuacg cugaagagau gcucgaugug
1080
guuaaucagg aacuguuggc ccuucacugc cacugaccgg augccgacgc gaagcgggua
1140
gauaucacac ucugucuggc uuuuggcugu gacgcacagu ucauagagau aaccuucacc
1200
cgguugccag aggugcggau ucaccacuug caaagucccg cuagugccuu guccaguugc
1260
aaccaccugu ugauccgcau cacgcaguuc aacgcugaca ucaccauugg ccaccaccug
1320
ccaguc
1326
<210> 3
<211> 1485
<212> РНК
<213> Искусственная последовательность
<220>
<223> FAD2.1 кпРНК
<400> 3
gaagaucugu agccucucgc ggucauugua gauugggccg uaagggucau agugacaugc
60
aaagcgauca uaaugucggc cagaaacauu gaaagccaag uacaaaggcc agccaagagu
120
aagggugauc guaagugaaa uaacccggcc ugguggauug uucaaguacu uggaauacca
180
uccgaguugu gauuucggcu uaggcacaaa aaccucaucg cgcucgagug agccaguguu
240
ggaguggugg cgacgaugac uauauuucca agagaaguag ggcaccauca gagcagagug
300
gaggauaagc ccgacagugu caucaaccca cugguaguca cuaaaggcau gguggccaca
360
uucgugcgca auaacccaaa uaccagugca aacacaaccc ugacaaaucc aguaaauagg
420
ccaugcaagg uagcaauccu aggcacucug cucugauggu gcccuacuuc ucuuggaaau
480
auagucaucg ucgccaccac uccaacacug gcucacucga gcgcgaugag guuuuugugc
540
cuaagccgaa aucacaacuc ggaugguauu ccaaguacuu gaacaaucca ccaggccggg
600
uuauuucacu uacgaucacc cuuacucuug gcuggccuuu guacuuggcu uucaauguuu
660
cuggccgaca uuaugaucgc uuugcauguc acuaugaccc uuacggccca aucuacaaug
720
accgcgagag gcuacagauc uuccuuucug augcuggagu uauuggagcu gguuaucuac
780
uauaucguau ugccuuggua aaagggcuag cuuggcucgu guguauguau ggcguaccac
840
uccuaaucgu gaacggcuuc cuugucuuga ucacuuauuu gcagcacacu cacccgucau
900
ugccucacua cgauucaucc gaaugggauu ggcuaagggg agcuuuggca accgucgaca
960
gagacuaugg cauucuaaac aaggucuucc acaacaucac cgauacucac guaguccacc
1020
aucuguucuc gaccaugcca cacucuagag ugaugcuuca ucuuucucca cauagauaca
1080
cucuuuugcu ucccuccaca uugccuugaa aaccgggguu ccgucaaauu gguaguaguc
1140
uccgaguaau ggcuugacug cuuuuguugc cuccauugca uuguagugug gcauggucga
1200
gaacagaugg uggacuacgu gaguaucggu gauguugugg aagaccuugu uuagaaugcc
1260
auagucucug ucgacgguug ccaaagcucc ccuuagccaa ucccauucgg augaaucgua
1320
gugaggcaau gacgggugag ugugcugcaa auaagugauc aagacaagga agccguucac
1380
gauuaggagu gguacgccau acauacacac gagccaagcu agcccuuuua ccaaggcaau
1440
acgauauagu agauaaccag cuccaauaac uccagcauca gaaag
1485
<210> 4
<211> 1258
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Конструкция ДНК, кодирующая GFP кпРНК
<400> 4
taatacgact cactataggg tgtcgccctc gaacttcacc tcggcgcggg tcttgtagtt
60
gccgtcgtcc ttgaagaaga tggtgcgctc ctggacgtag ccttcgggca tggcggactt
120
gaagaagtcg tgctgcttca tgtggtcggg gtagcggctg aagcactgca cgccgtaggt
180
gaaggtggtc acgagggtgg gccagggcac gggcagcttg ccggtggtgc agatgaactt
240
cagggtcagc ttgccgtagg tggcatcgcc ctcgccctcg ccggacacgc tgaacttgtg
300
gccgtttacg tcgccgtcca gctcgaccag gatgggcacc accccggtga acagctcctc
360
gcccttgctc actatggatc aactagggat ccccctgaag ttcatctgca ccaccggcaa
420
gctgcccgtg ccctggccca ccctcgtgac caccttcacc tacggcgtgc agtgcttcag
480
ccgctacccc gaccacatga agcagcacga cttcttcaag tccgccatgc ccgaaggcta
540
cgtccaggag cgcaccatct tcttcaagga cgacggcaac tacaagaccc gcgccgaggt
600
gaagttcgag ggcgacaccc tggtgaaccg catcgagctg aagggcatcg acttcaagga
660
ggacggcaac atcctggggc acaagctgga gtacaactac aacagccaca acgtctatat
720
catggccgac aagcagaaga acggcatcaa ggtgaacttc aagatccgcc acaacatcga
780
ggacggcagc gtgcagctcg ccgaccacta ccagcagaac acccccatcg gcgacggccc
840
cgtgctgctg ccaagcttta ggtgatccaa gcttgatccg ggctttactt gtacagctcg
900
tccatgccga gagtgatccc ggcggcggtc acgaactcca gcaggaccat gtgatcgcgc
960
ttctcgttgg ggtctttgct cagggcggac tgggtgctca ggtagtggtt gtcgggcagc
1020
agcacggggc cgtcgccgat gggggtgttc tgctggtagt ggtcggcgag ctgcacgctg
1080
ccgtcctcga tgttgtggcg gatcttgaag ttcaccttga tgccgttctt ctgcttgtcg
1140
gccatgatat agacgttgtg gctgttgtag ttgtactcca gcttgtgccc caggatgttg
1200
ccgtcctcct tgaagtcgat gcccttcagc tcgatgcggt tcaccattgt cgggatac
1258
<210> 5
<211> 1346
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Конструкция ДНК, кодирующая Gus кпРНК
<400> 5
taatacgact cactataggg aacagacgcg tggttacagt cttgcgcgac atgcgtcacc
60
acggtgatat cgtccaccca ggtgttcggc gtggtgtaga gcattacgct gcgatggatt
120
ccggcatagt taaagaaatc atggaagtaa gactgctttt tcttgccgtt ttcgtcggta
180
atcaccattc ccggcgggat agtctgccag ttcagttcgt tgttcacaca aacggtgata
240
cgtacacttt tcccggcaat aacatacggc gtgacatcgg cttcaaatgg cgtatagccg
300
ccctgatgct ccatcacttc ctgattattg acccacactt tgccgtaatg agtgaccgca
360
tcgaaacgca gcacgatacg ctggcctgcc caacctttcg gtataaagac ttcgcgctga
420
taccagacgt gccgtatgtt attgccggga aaagtgtacg tatcaccgtt tgtgtgaaca
480
acgaactgaa ctggcagact atcccgccgg gaatggtgat taccgacgaa aacggcaaga
540
aaaagcagtc ttacttccat gatttcttta actatgccgg aatccatcgc agcgtaatgc
600
tctacaccac gccgaacacc tgggtggacg atatcaccgt ggtgacgcat gtcgcgcaag
660
actgtaacca cgcgtctgtt cccgactggc aggtggtggc caatggtgat gtcagcgttg
720
aactgcgtga tgcggatcaa caggtggttg caactggaca aggcactagc gggactttgc
780
aagtggtgaa tccgcacctc tggcaaccgg gtgaaggtta tctctatgaa ctgtgcgtca
840
cagccaaaag ccagacagag tgtgatatct acccgcttcg cgtcggcatc cggtcagtgg
900
cagtgaaggg ccaacagttc ctgattaacc acaaaccgtt ctactttact ggctttggtc
960
gtcatgaaga tgcggactta cgtggcaaag gattcgataa cgtgctgatg gtgcacgacc
1020
acgcattaat ggactggatt ggggccaact cctaccgtac ctcgcattac ccttacgctg
1080
aagagatgct cgatgtggtt aatcaggaac tgttggccct tcactgccac tgaccggatg
1140
ccgacgcgaa gcgggtagat atcacactct gtctggcttt tggctgtgac gcacagttca
1200
tagagataac cttcacccgg ttgccagagg tgcggattca ccacttgcaa agtcccgcta
1260
gtgccttgtc cagttgcaac cacctgttga tccgcatcac gcagttcaac gctgacatca
1320
ccattggcca ccacctgcca gtcaac
1346
<210> 6
<211> 1512
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Конструкция ДНК, кодирующая FAD2.1 кпРНК
<400> 6
atttaggtga cactatagaa gatctgtagc ctctcgcggt cattgtagat tgggccgtaa
60
gggtcatagt gacatgcaaa gcgatcataa tgtcggccag aaacattgaa agccaagtac
120
aaaggccagc caagagtaag ggtgatcgta agtgaaataa cccggcctgg tggattgttc
180
aagtacttgg aataccatcc gagttgtgat ttcggcttag gcacaaaaac ctcatcgcgc
240
tcgagtgagc cagtgttgga gtggtggcga cgatgactat atttccaaga gaagtagggc
300
accatcagag cagagtggag gataagcccg acagtgtcat caacccactg gtagtcacta
360
aaggcatggt ggccacattc gtgcgcaata acccaaatac cagtgcaaac acaaccctga
420
caaatccagt aaataggcca tgcaaggtag caatcctagg cactctgctc tgatggtgcc
480
ctacttctct tggaaatata gtcatcgtcg ccaccactcc aacactggct cactcgagcg
540
cgatgaggtt tttgtgccta agccgaaatc acaactcgga tggtattcca agtacttgaa
600
caatccacca ggccgggtta tttcacttac gatcaccctt actcttggct ggcctttgta
660
cttggctttc aatgtttctg gccgacatta tgatcgcttt gcatgtcact atgaccctta
720
cggcccaatc tacaatgacc gcgagaggct acagatcttc ctttctgatg ctggagttat
780
tggagctggt tatctactat atcgtattgc cttggtaaaa gggctagctt ggctcgtgtg
840
tatgtatggc gtaccactcc taatcgtgaa cggcttcctt gtcttgatca cttatttgca
900
gcacactcac ccgtcattgc ctcactacga ttcatccgaa tgggattggc taaggggagc
960
tttggcaacc gtcgacagag actatggcat tctaaacaag gtcttccaca acatcaccga
1020
tactcacgta gtccaccatc tgttctcgac catgccacac tctagagtga tgcttcatct
1080
ttctccacat agatacactc ttttgcttcc ctccacattg ccttgaaaac cggggttccg
1140
tcaaattggt agtagtctcc gagtaatggc ttgactgctt ttgttgcctc cattgcattg
1200
tagtgtggca tggtcgagaa cagatggtgg actacgtgag tatcggtgat gttgtggaag
1260
accttgttta gaatgccata gtctctgtcg acggttgcca aagctcccct tagccaatcc
1320
cattcggatg aatcgtagtg aggcaatgac gggtgagtgt gctgcaaata agtgatcaag
1380
acaaggaagc cgttcacgat taggagtggt acgccataca tacacacgag ccaagctagc
1440
ccttttacca aggcaatacg atatagtaga taaccagctc caataactcc agcatcagaa
1500
agcccgggac tc
1512
<210> 7
<211> 732
<212> ДНК
<213> Aequorea victoria
<400> 7
atggtgagca agggcgagga gctgttcacc ggggtggtgc ccatcctggt cgagctggac
60
ggcgacgtaa acggccacaa gttcagcgtg tccggcgagg gcgagggcga tgccacctac
120
ggcaagctga ccctgaagtt catctgcacc accggcaagc tgcccgtgcc ctggcccacc
180
ctcgtgacca ccttcaccta cggcgtgcag tgcttcagcc gctaccccga ccacatgaag
240
cagcacgact tcttcaagtc cgccatgccc gaaggctacg tccaggagcg caccatcttc
300
ttcaaggacg acggcaacta caagacccgc gccgaggtga agttcgaggg cgacaccctg
360
gtgaaccgca tcgagctgaa gggcatcgac ttcaaggagg acggcaacat cctggggcac
420
aagctggagt acaactacaa cagccacaac gtctatatca tggccgacaa gcagaagaac
480
ggcatcaagg tgaacttcaa gatccgccac aacatcgagg acggcagcgt gcagctcgcc
540
gaccactacc agcagaacac ccccatcggc gacggccccg tgctgctgcc cgacaaccac
600
tacctgagca cccagtccgc cctgagcaaa gaccccaacg agaagcgcga tcacatggtc
660
ctgctggagt tcgtgaccgc cgccgggatc actctcggca tggacgagct gtacaagtaa
720
agcccggatc tc
732
<210> 8
<211> 1855
<212> ДНК
<213> Escherichia coli
<400> 8
atggtccgtc ctgtagaaac cccaacccgt gaaatcaaaa aactcgacgg cctgtgggca
60
ttcagtctgg atcgcgaaaa ctgtggaatt gatcagcgtt ggtgggaaag cgcgttacaa
120
gaaagccggg caattgctgt gccaggcagt tttaacgatc agttcgccga tgcagatatt
180
cgtaattatg cgggcaacgt ctggtatcag cgcgaagtct ttataccgaa aggttgggca
240
ggccagcgta tcgtgctgcg tttcgatgcg gtcactcatt acggcaaagt gtgggtcaat
300
aatcaggaag tgatggagca tcagggcggc tatacgccat ttgaagccga tgtcacgccg
360
tatgttattg ccgggaaaag tgtacgtatc accgtttgtg tgaacaacga actgaactgg
420
cagactatcc cgccgggaat ggtgattacc gacgaaaacg gcaagaaaaa gcagtcttac
480
ttccatgatt tctttaacta tgccggaatc catcgcagcg taatgctcta caccacgccg
540
aacacctggg tggacgatat caccgtggtg acgcatgtcg cgcaagactg taaccacgcg
600
tctgttcccg actggcaggt ggtggccaat ggtgatgtca gcgttgaact gcgtgatgcg
660
gatcaacagg tggttgcaac tggacaaggc actagcggga ctttgcaagt ggtgaatccg
720
cacctctggc aaccgggtga aggttatctc tatgaactgt gcgtcacagc caaaagccag
780
acagagtgtg atatctaccc gcttcgcgtc ggcatccggt cagtggcagt gaagggccaa
840
cagttcctga ttaaccacaa accgttctac tttactggct ttggtcgtca tgaagatgcg
900
gacttacgtg gcaaaggatt cgataacgtg ctgatggtgc acgaccacgc attaatggac
960
tggattgggg ccaactccta ccgtacctcg cattaccctt acgctgaaga gatgctcgac
1020
tgggcagatg aacatggcat cgtggtgatt gatgaaactg ctgctgtcgg ctttaacctc
1080
tctttaggca ttggtttcga agcgggcaac aagccgaaag aactgtacag cgaagaggca
1140
gtcaacgggg aaactcagca agcgcactta caggcgatta aagagctgat agcgcgtgac
1200
aaaaaccacc caagcgtggt gatgtggagt attgccaacg aaccggatac ccgtccgcaa
1260
gtgcacggga atatttcgcc actggcggaa gcaacgcgta aactcgaccc gacgcgtccg
1320
atcacctgcg tcaatgtaat gttctgcgac gctcacaccg ataccatcag cgatctcttt
1380
gatgtgctgt gcctgaaccg ttattacgga tggtatgtcc aaagcggcga tttggaaacg
1440
gcagagaagg tactggaaaa agaacttctg gcctggcagg agaaactgca tcagccgatt
1500
atcatcaccg aatacggcgt ggatacgtta gccgggctgc actcaatgta caccgacatg
1560
tggagtgaag agtatcagtg tgcatggctg gatatgtatc accgcgtctt tgatcgcgtc
1620
agcgccgtcg tcggtgaaca ggtatggaat ttcgccgatt ttgcgacctc gcaaggcata
1680
ttgcgcgttg gcggtaacaa gaaagggatc ttcactcgcg accgcaaacc gaagtcggcg
1740
gcttttctgc tgcaaaaacg ctggactggc atgaacttcg gtgaaaaacc gcagcaggga
1800
ggcaaacaat gaatcaacaa ctctcctggc gcaccatcgt cggctacagc ctcgg
1855
<210> 9
<211> 1152
<212> ДНК
<213> Nicotiana benthamiana
<400> 9
atgggagctg gtggtaatat gtctcttgta accagcaaga ctggcgaaaa gaagaatcct
60
cttgaaaagg taccaacctc aaagcctcct ttcacagttg gtgatatcaa gaaggccatc
120
ccacctcact gctttcagcg gtctctcgtt cgttcgttct cctatgttgt gtatgacctt
180
ttactggtgt ccgtcttcta ctacattgcc accacttact tccacctcct cccgtcccca
240
tattgctacc ttgcatggcc tatttactgg atttgtcagg gttgtgtttg cactggtatt
300
tgggttattg cgcacgaatg tggccaccat gcctttagtg actaccagtg ggttgatgac
360
actgtcgggc ttatcctcca ctctgctctg atggtgccct acttctcttg gaaatatagt
420
catcgtcgcc accactccaa cactggctca ctcgagcgcg atgaggtttt tgtgcctaag
480
ccgaaatcac aactcggatg gtattccaag tacttgaaca atccaccagg ccgggttatt
540
tcacttacga tcacccttac tcttggctgg cctttgtact tggctttcaa tgtttctggc
600
cgacattatg atcgctttgc atgtcactat gacccttacg gcccaatcta caatgaccgc
660
gagaggctac agatcttcct ttctgatgct ggagttattg gagctggtta tctactatat
720
cgtattgcct tggtaaaagg gctagcttgg ctcgtgtgta tgtatggcgt accactccta
780
atcgtgaacg gcttccttgt cttgatcact tatttgcagc acactcaccc gtcattgcct
840
cactacgatt catccgaatg ggattggcta aggggagctt tggcaaccgt cgacagagac
900
tatggcattc taaacaaggt cttccacaac atcaccgata ctcacgtagt ccaccatctg
960
ttctcgacca tgccacacta caatgcaatg gaggcaacaa aagcagtcaa gccattactc
1020
ggagactact accaatttga cggaaccccg gttttcaagg caatgtggag ggaagcaaaa
1080
gagtgtatct atgtggagaa agatgaagca tcacaaggca aaggtgtttt ctggtacaaa
1140
aacaaattct ga
1152
<210> 10
<211> 225
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Смысловая область GUS для конструкций, кодирующих молекулы
шпилечной РНК
направленные на мРНК GUS
<400> 10
cctcgaggat cctcgcgtcg gcatccggtc agtggcagtg aagggcgaac agttcctgat
60
taaccacaaa ccgttctact ttactggctt tggtcgtcat gaagatgcgg acttgcgtgg
120
caaaggattc gataacgtgc tgatggtgca cgaccacgca ttaatggact ggattggggc
180
caactcctac cgtacctcgc attaccctta cgaagcttgg taccc
225
<210> 11
<211> 216
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Смысловая область GUS для конструкции, кодирующей молекулу
шпилечной
РНК hpGUS[G:U]
<400> 11
ccctcgagtt gtgttggtat ttggttagtg gtagtgaagg gtgaatagtt tttgattaat
60
tataaattgt tttattttat tggttttggt tgttatgaag atgtggattt gtgtggtaaa
120
ggatttgata atgtgttgat ggtgtatgat tatgtattaa tggattggat tggggttaat
180
ttttattgta ttttgtatta tttttatggg tacccc
216
<210> 12
<211> 216
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Смысловая область GUS для конструкций, кодирующих молекулу
шпилечной
РНК hpGUS[1:4]
<400> 12
ccctcgagtc gggtccgcaa ccgctcactg ggagtcaagc gcgtacactt cgtgaataag
60
cactaacggt tgtacattag tgggtttcgt cctcaagaac atggggagtt gggtgccaat
120
ggaatcgtta aggtggtgaa ggtccaccac ctcgctttat tggtctgcat tcggggcaag
180
tccaacccta cgtcggattt cccataccgg tacccc
216
<210> 13
<211> 216
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Смысловая область GUS для конструкций, кодирующих молекулу
шпилечной
РНК hpGUS[2:10]
<400> 13
ccctcgagtc gcgtcgcgat ccggtctctg gcagtgttgg gcgaactctt cctgatatac
60
cacaaagggt tctactaaac tggcttacgt cgtcatctag atgcggtgtt gcgtgggtaa
120
ggattcctta acgtgcacat ggtgcagcac cacgcaaaaa tggactccat tggggcgtac
180
tcctacgcta cctcgctata cccttagcgg tacccc
216
<210> 14
<211> 240
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Последовательность ДНК нуклеотидов 781-1020 кодирующей белок
области
гена GUS
<400> 14
gagtgtgata tctacccgct tcgcgtcggc atccggtcag tggcagtgaa gggcgaacag
60
ttcctgatta accacaaacc gttctacttt actggctttg gtcgtcatga agatgcggac
120
ttgcgtggca aaggattcga taacgtgctg atggtgcacg accacgcatt aatggactgg
180
attggggcca actcctaccg tacctcgcat tacccttacg ctgaagagat gctcgactgg
240
<210> 15
<211> 463
<212> РНК
<213> Искусственная последовательность
<220>
<223> шпилечная структура (включающая петлю) РНК hpGUS[wt]
<400> 15
ggauccucgc gucggcaucc ggucaguggc agugaagggc gaacaguucc ugauuaacca
60
caaaccguuc uacuuuacug gcuuuggucg ucaugaagau gcggacuugc guggcaaagg
120
auucgauaac gugcugaugg ugcacgacca cgcauuaaug gacuggauug gggccaacuc
180
cuaccguacc ucgcauuacc cuuacgaagc uugguacccc agcuuguugg gaagcugggu
240
ucgaaaucga uaagcuucgu aaggguaaug cgagguacgg uaggaguugg ccccaaucca
300
guccauuaau gcguggucgu gcaccaucag cacguuaucg aauccuuugc cacgcaaguc
360
cgcaucuuca ugacgaccaa agccaguaaa guagaacggu uugugguuaa ucaggaacug
420
uucgcccuuc acugccacug accggaugcc gacgcgagga ucc
463
<210> 16
<211> 457
<212> РНК
<213> Искусственная последовательность
<220>
<223> шпилечная структура (включающая петлю) РНК hpGUS[G:U]
<400> 16
cucgaguugu guugguauuu gguuaguggu agugaagggu gaauaguuuu ugauuaauua
60
uaaauuguuu uauuuuauug guuuugguug uuaugaagau guggauuugu gugguaaagg
120
auuugauaau guguugaugg uguaugauua uguauuaaug gauuggauug ggguuaauuu
180
uuauuguauu uuguauuauu uuuaugggua ccccagcuug uugggaagcu ggguucgaaa
240
ucgauaagcu ucguaagggu aaugcgaggu acgguaggag uuggccccaa uccaguccau
300
uaaugcgugg ucgugcacca ucagcacguu aucgaauccu uugccacgca aguccgcauc
360
uucaugacga ccaaagccag uaaaguagaa cgguuugugg uuaaucagga acuguucgcc
420
cuucacugcc acugaccgga ugccgacgcg aggaucc
457
<210> 17
<211> 457
<212> РНК
<213> Искусственная последовательность
<220>
<223> шпилечная структура (включающая петлю) РНК hpGUS[1:4]
<400> 17
cucgagucgg guccgcaacc gcucacuggg agucaagcgc guacacuucg ugaauaagca
60
cuaacgguug uacauuagug gguuucgucc ucaagaacau ggggaguugg gugccaaugg
120
aaucguuaag guggugaagg uccaccaccu cgcuuuauug gucugcauuc ggggcaaguc
180
caacccuacg ucggauuucc cauaccggua ccccagcuug uugggaagcu ggguucgaaa
240
ucgauaagcu ucguaagggu aaugcgaggu acgguaggag uuggccccaa uccaguccau
300
uaaugcgugg ucgugcacca ucagcacguu aucgaauccu uugccacgca aguccgcauc
360
uucaugacga ccaaagccag uaaaguagaa cgguuugugg uuaaucagga acuguucgcc
420
cuucacugcc acugaccgga ugccgacgcg aggaucc
457
<210> 18
<211> 457
<212> РНК
<213> Искусственная последовательность
<220>
<223> шпилечная структура (включающая петлю) РНК hpGUS[2:10]
<400> 18
cucgagucgc gucgcgaucc ggucucuggc aguguugggc gaacucuucc ugauauacca
60
caaaggguuc uacuaaacug gcuuacgucg ucaucuagau gcgguguugc guggguaagg
120
auuccuuaac gugcacaugg ugcagcacca cgcaaaaaug gacuccauug gggcguacuc
180
cuacgcuacc ucgcuauacc cuuagcggua ccccagcuug uugggaagcu ggguucgaaa
240
ucgauaagcu ucguaagggu aaugcgaggu acgguaggag uuggccccaa uccaguccau
300
uaaugcgugg ucgugcacca ucagcacguu aucgaauccu uugccacgca aguccgcauc
360
uucaugacga ccaaagccag uaaaguagaa cgguuugugg uuaaucagga acuguucgcc
420
cuucacugcc acugaccgga ugccgacgcg aggaucc
457
<210> 19
<211> 4851
<212> ДНК
<213> Arabidopsis thaliana
<400> 19
atctctctct ttcgatggaa ctgagctctt tctctctttc ctcttctttt ctctctctat
60
ctctatctct cgtagcttga taagagtttc tctcttttga agatccgttt ctctctctct
120
cactgagact attgttgtta ggtcaacttg cgatcatggc gatttcgaag gtctgaagct
180
gatttcgaat ggtttggaga tatccgtagt ggttaagcat atggaagtct atgttctgct
240
cttggttgct ctgttagggc ttcctccatt tggaccaact tagctgaatg ttgtatgatc
300
tctctccttg aagcagcaaa taagaagaag gtctggtcct taacttaaca tctggttact
360
agaggaaact tcagctatta ttaggtaaag aaagactgta cagagttgta taacaagtaa
420
gcgttagagt ggctttgttt gcctcggtga tagaagaacc gactgattcg ttgttgtgtg
480
ttagctttgg agggaatcag atttcgcgag ggaaggtgtt ttagatcaaa tctgtgaatt
540
ttactcaact gaggctttta gtgaaccacg actgtagagt tgaccttgaa tcctactctg
600
agtaattata ttatcagata gatttaggat ggaagctgaa attgtgaatg tgagacctca
660
gctagggttt atccagagaa tggttcctgc tctacttcct gtccttttgg tttctgtcgg
720
atatattgat cccgggaaat gggttgcaaa tatcgaagga ggtgctcgtt tcgggtatga
780
cttggtggca attactctgc ttttcaattt tgccgccatc ttatgccaat atgttgcagc
840
tcgcataagc gttgtgactg gtaaacactt ggctcagatc tgcaatgaag aatatgacaa
900
gtggacgtgc atgttcttgg gcattcaggc ggagttctca gcaattctgc tcgaccttac
960
catggttgtg ggagttgcgc atgcacttaa ccttttgttt ggggtggagt tatccactgg
1020
agtgtttttg gccgccatgg atgcgttttt atttcctgtt ttcgcctctt tccttgaaaa
1080
tggtatggca aatacagtat ccatttactc tgcaggcctg gtattacttc tctatgtatc
1140
tggcgtcttg ctgagtcagt ctgagatccc actctctatg aatggagtgt taactcggtt
1200
aaatggagag agcgcattcg cactgatggg tcttcttggc gcaagcatcg tccctcacaa
1260
tttttatatc cattcttatt ttgctgggga aagtacatct tcgtctgatg tcgacaagag
1320
cagcttgtgt caagaccatt tgttcgccat ctttggtgtc ttcagcggac tgtcacttgt
1380
aaattatgta ttgatgaatg cagcagctaa tgtgtttcac agtactggcc ttgtggtact
1440
gacttttcac gatgccttgt cactaatgga gcaggtattt atgagtccgc tcattccagt
1500
ggtctttttg atgctcttgt tcttctctag tcaaattacc gcactagctt gggctttcgg
1560
tggagaggtc gtcctgcatg acttcctgaa gatagaaata cccgcttggc ttcatcgtgc
1620
tacaatcaga attcttgcag ttgctcctgc gctttattgt gtatggacat ctggtgcaga
1680
cggaatatac cagttactta tattcaccca ggtcttggtg gcaatgatgc ttccttgctc
1740
ggtaataccg cttttccgca ttgcttcgtc gagacaaatc atgggtgtcc ataaaatccc
1800
tcaggttggc gagttcctcg cacttacaac gtttttggga tttctggggt tgaatgttgt
1860
ttttgttgtt gagatggtat ttgggagcag tgactgggct ggtggtttga gatggaatac
1920
cgtgatgggc acctcgattc agtacaccac tctgcttgta tcgtcatgtg catccttatg
1980
cctgatactc tggctggcag ccacgccgct gaaatctgcg agtaacagag cggaagctca
2040
aatatggaac atggatgctc aaaatgcttt atcttatcca tctgttcaag aagaggaaat
2100
tgaaagaaca gaaacaagga ggaacgaaga cgaatcaata gtgcggttgg aaagcagggt
2160
aaaggatcag ttggatacta cgtctgttac tagctcggtc tatgatttgc cagagaacat
2220
tctaatgacg gatcaagaaa tccgttcgag ccctccagag gaaagagagt tggatgtaaa
2280
gtactctacc tctcaagtta gtagtcttaa ggaagactct gatgtaaagg aacagtctgt
2340
attgcagtca acagtggtta atgaggtcag tgataaggat ctgattgttg aaacaaagat
2400
ggcgaaaatt gaaccaatga gtcctgtgga gaagattgtt agcatggaga ataacagcaa
2460
gtttattgaa aaggatgttg aaggggtttc atgggaaaca gaagaagcta ccaaagctgc
2520
tcctacaagc aactttactg tcggatctga tggtcctcct tcattccgca gcttaagtgg
2580
ggaaggggga agtgggactg gaagcctttc acggttgcaa ggtttgggac gtgctgcccg
2640
gagacactta tctgcgatcc ttgatgaatt ttggggacat ttatatgatt ttcatgggca
2700
attggttgct gaagccaggg caaagaaact agatcagctg tttggcactg atcaaaagtc
2760
agcctcttct atgaaagcag attcgtttgg aaaagacatt agcagtggat attgcatgtc
2820
accaactgcg aagggaatgg attcacagat gacttcaagt ttatatgatt cactgaagca
2880
gcagaggaca ccgggaagta tcgattcgtt gtatggatta caaagaggtt cgtcaccgtc
2940
accgttggtc aaccgtatgc agatgttggg tgcatatggt aacaccacta ataataataa
3000
tgcttacgaa ttgagtgaga gaagatactc tagcctgcgt gctccatcat cttcagaggg
3060
ttgggaacac caacaaccag ctacagttca cggataccag atgaagtcat atgtagacaa
3120
tttggcaaaa gaaaggcttg aagccttaca atcccgtgga gagatcccga catcgagatc
3180
tatggcgctt ggtacattga gctatacaca gcaacttgct ttagccttga aacagaagtc
3240
ccagaatggt ctaacccctg gaccagctcc tgggtttgag aattttgctg ggtctagaag
3300
catatcgcga caatctgaaa gatcttatta cggtgttcca tcttctggca atactgatac
3360
tgttggcgca gcagtagcca atgagaaaaa atatagtagc atgccagata tctcaggatt
3420
gtctatgtcc gcaaggaaca tgcatttacc aaacaacaag agtggatact gggatccgtc
3480
aagtggagga ggagggtatg gtgcgtctta tggtcggtta agcaatgaat catcgttata
3540
ttctaatttg gggtcacggg tgggagtacc ctcgacttat gatgacattt ctcaatcaag
3600
aggaggctac agagatgcct acagtttgcc acagagtgca acaacaggga ccggatcgct
3660
ttggtccaga cagccctttg agcagtttgg tgtagcggag aggaatggtg ctgttggtga
3720
ggagctcagg aatagatcga atccgatcaa tatagacaac aacgcttctt ctaatgttga
3780
tgcagaggct aagcttcttc agtcgttcag gcactgtatt ctaaagctta ttaaacttga
3840
aggatccgag tggttgtttg gacaaagcga tggagttgat gaagaactga ttgaccgggt
3900
agctgcacga gagaagttta tctatgaagc tgaagctcga gaaataaacc aggtgggtca
3960
catgggggag ccactaattt catcggttcc taactgtgga gatggttgcg tttggagagc
4020
tgatttgatt gtgagctttg gagtttggtg cattcaccgt gtccttgact tgtctctcat
4080
ggagagtcgg cctgagcttt ggggaaagta cacttacgtt ctcaaccgcc tacagggagt
4140
gattgatccg gcgttctcaa agctgcggac accaatgaca ccgtgctttt gccttcagat
4200
tccagcgagc caccagagag cgagtccgac ttcagctaac ggaatgttac ctccggctgc
4260
aaaaccggct aaaggcaaat gcacaaccgc agtcacactt cttgatctaa tcaaagacgt
4320
tgaaatggca atctcttgta gaaaaggccg aaccggtaca gctgcaggtg atgtggcttt
4380
cccaaagggg aaagagaatt tggcttcggt tttgaagcgg tataaacgtc ggttatcgaa
4440
taaaccagta ggtatgaatc aggatggacc cggttcaaga aaaaacgtga ctgcgtacgg
4500
atcattgggt tgaagaagaa gaacattgtg agaaatctca tgatcaaagt gacgtcgaga
4560
gggaagccga agaatcaaaa ctctcgcttt tgattgctcc tctgcttcgt taattgtgta
4620
ttaagaaaag aagaaaaaaa atggattttt gttgcttcag aatttttcgc tctttttttc
4680
ttaatttggt tgtaatgtta tgtttatata catatatcat catcatagga ccatagctac
4740
aaaccgaatc cggtttgtgt aattctatgc ggaatcataa agaaatcgtc ggtttgaaat
4800
gttaaatctc ctaaaccgga tctctgcacg tagctgacac atcgacgcta g
4851
<210> 20
<211> 1703
<212> ДНК
<213> Arabidopsis thaliana
<400> 20
gttgcaaata tataaatcaa tcaaaagatt taaaacccac cattcaatct tggtaagtaa
60
cgaaaaaaaa gggaagcaag aagaaccaca gaaaaggggg ctaacaacta gacacgtaga
120
tcttcatctg cccgtccatc taacctacca cactctcatc ttctttttcc cgtgtcagtt
180
tgttatataa gctctcactc tccggtatat ttccaaatac acctaacttg tttagtacac
240
aacagcaaca tcaaactcta ataaacccaa gttggtgtat actataatgg tgatggctgg
300
tgcttcttct ttggatgaga tcagacaggc tcagagagct gatggacctg caggcatctt
360
ggctattggc actgctaacc ctgagaacca tgtgcttcag gcggagtatc ctgactacta
420
cttccgcatc accaacagtg aacacatgac cgacctcaag gagaagttca agcgcatgtg
480
cgacaagtcg acaattcgga aacgtcacat gcatctgacg gaggaattcc tcaaggaaaa
540
cccacacatg tgtgcttaca tggctccttc tctggacacc agacaggaca tcgtggtggt
600
cgaagtccct aagctaggca aagaagcggc agtgaaggcc atcaaggagt ggggccagcc
660
caagtcaaag atcactcatg tcgtcttctg cactacctcc ggcgtcgaca tgcctggtgc
720
tgactaccag ctcaccaagc ttcttggtct ccgtccttcc gtcaagcgtc tcatgatgta
780
ccagcaaggt tgcttcgccg gcggtactgt cctccgtatc gctaaggatc tcgccgagaa
840
caatcgtgga gcacgtgtcc tcgttgtctg ctctgagatc acagccgtta ccttccgtgg
900
tccctctgac acccaccttg actccctcgt cggtcaggct cttttcagtg atggcgccgc
960
cgcactcatt gtggggtcgg accctgacac atctgtcgga gagaaaccca tctttgagat
1020
ggtgtctgcc gctcagacca tccttccaga ctctgatggt gccatagacg gacatttgag
1080
ggaagttggt ctcaccttcc atctcctcaa ggatgttccc ggcctcatct ccaagaacat
1140
tgtgaagagt ctagacgaag cgtttaaacc tttggggata agtgactgga actccctctt
1200
ctggatagcc caccctggag gtccagcgat cctagaccag gtggagataa agctaggact
1260
aaaggaagag aagatgaggg cgacacgtca cgtgttgagc gagtatggaa acatgtcgag
1320
cgcgtgcgtt ctcttcatac tagacgagat gaggaggaag tcagctaagg atggtgtggc
1380
cacgacagga gaagggttgg agtggggtgt cttgtttggt ttcggaccag gtctcactgt
1440
tgagacagtc gtcttgcaca gcgttcctct ctaaacagaa cgcttgcctt ctatctgcct
1500
acctacctac gcaaaacttt aatcctgtct tatgttttat ataatataat cattatatgt
1560
ttacgcaata attaaggaag aattcatttg atgtgatatg tgatatgtgc tggacaggtc
1620
tattcgactg tttttgtact ctcttttttg tgtcttttta caatattaaa tctatgggtc
1680
ttgaatgaca tcaaatcttt gtt
1703
<210> 21
<211> 229
<212> ДНК
<213> Arabidopsis thaliana
<400> 21
cctcgaggat cctctagacc tcagctaggg tttatccaga gaatggttcc tgctctactt
60
cctgtccttt tggtttctgt cggatatatt gatcccggga aatgggttgc aaatatcgaa
120
ggaggtgctc gtttcgggta tgacttggtg gcaattactc tgcttttcaa ttttgccgcc
180
atcttatgcc aatatgttgc agctcgcata agcgttaagc ttggtaccc
229
<210> 22
<211> 218
<212> ДНК
<213> Искусственная последовательность
<220>
<223> фрагмент, включающий 200 нт смысловую последовательность EIN2
<400> 22
cctcgagtct agattttagt tagggtttat ttagagaatg gtttttgttt tattttttgt
60
ttttttggtt tttgttggat atattgattt tgggaaatgg gttgtaaata ttgaaggagg
120
tgtttgtttt gggtatgatt tggtggtaat tattttgttt tttaattttg ttgttatttt
180
atgttaatat gttgtagttt gtataagtgt tggtaccc
218
<210> 23
<211> 230
<212> ДНК
<213> Arabidopsis thaliana
<400> 23
cctcgaggat ccgttgtctg ctctgagatc acagccgtta ccttccgtgg tccctctgac
60
acccaccttg actccctcgt cggtcaggct cttttcagtg atggcgccgc cgcactcatt
120
gtggggtcgg accctgacac atctgtcgga gagaaaccca tctttgagat ggtgtctgcc
180
gctcagacca tccttccaga ctctgatggt gctctagaag cttggtaccc
230
<210> 24
<211> 219
<212> ДНК
<213> Искусственная последовательность
<220>
<223> фрагмент, включающий 200 нт смысловую последовательность CHS
<400> 24
cctcgaggtt gtttgttttg agattatagt tgttattttt tgtggttttt ttgatattta
60
ttttgatttt tttgttggtt aggttttttt tagtgatggt gttgttgtat ttattgtggg
120
gttggatttt gatatatttg ttggagagaa atttattttt gagatggtgt ttgttgttta
180
gattattttt ttagattttg atggtgtcta gaggtaccc
219
<210> 25
<211> 219
<212> ДНК
<213> Искусственная последовательность
<220>
<223> фрагмент, включающий 200 нт антисмысловую последовательность
EIN2
<400> 25
caagcttaat gtttatgtga gttgtaatat attggtataa gatggtggta aaattgaaaa
60
gtagagtaat tgttattaag ttatatttga aatgagtatt ttttttgata tttgtaattt
120
attttttggg attaatatat ttgatagaaa ttaaaaggat aggaagtaga gtaggaatta
180
ttttttggat aaattttagt tgaggttcta gaggatccc
219
<210> 26
<211> 219
<212> ДНК
<213> Искусственная последовательность
<220>
<223> фрагмент, включающий 200 нт антисмысловую последовательность
CHS
<400> 26
caagcttcta gagtattatt agagtttgga aggatggttt gagtggtaga tattatttta
60
aagatgggtt tttttttgat agatgtgtta gggtttgatt ttataatgag tgtggtggtg
120
ttattattga aaagagtttg attgatgagg gagttaaggt gggtgttaga gggattatgg
180
aaggtaatgg ttgtgatttt agagtagata atggatccc
219
<210> 27
<211> 300
<212> ДНК
<213> Arabidopsis thaliana
<400> 27
agtaattata ttatcagata gatttaggat ggaagctgaa attgtgaatg tgagacctca
60
gctagggttt atccagagaa tggttcctgc tctacttcct gtccttttgg tttctgtcgg
120
atatattgat cccgggaaat gggttgcaaa tatcgaagga ggtgctcgtt tcgggtatga
180
cttggtggca attactctgc ttttcaattt tgccgccatc ttatgccaat atgttgcagc
240
tcgcataagc gttgtgactg gtaaacactt ggctcagatc tgcaatgaag aatatgacaa
300
<210> 28
<211> 300
<212> ДНК
<213> Arabidopsis thaliana
<400> 28
tccgtatcgc taaggatctc gccgagaaca atcgtggagc acgtgtcctc gttgtctgct
60
ctgagatcac agccgttacc ttccgtggtc cctctgacac ccaccttgac tccctcgtcg
120
gtcaggctct tttcagtgat ggcgccgccg cactcattgt ggggtcggac cctgacacat
180
ctgtcggaga gaaacccatc tttgagatgg tgtctgccgc tcagaccatc cttccagact
240
ctgatggtgc catagacgga catttgaggg aagttggtct caccttccat ctcctcaagg
300
<210> 29
<211> 240
<212> ДНК
<213> Arabidopsis thaliana
<400> 29
tcttcattgc agatctgagc caagtgttta ccagtcacaa cgcttatgcg agctgcaaca
60
tattggcata agatggcggc aaaattgaaa agcagagtaa ttgccaccaa gtcatacccg
120
aaacgagcac ctccttcgat atttgcaacc catttcccgg gatcaatata tccgacagaa
180
accaaaagga caggaagtag agcaggaacc attctctgga taaaccctag ctgaggtctc
240
<210> 30
<211> 300
<212> ДНК
<213> Arabidopsis thaliana
<400> 30
gatggaaggt gagaccaact tccctcaaat gtccgtctat ggcaccatca gagtctggaa
60
ggatggtctg agcggcagac accatctcaa agatgggttt ctctccgaca gatgtgtcag
120
ggtccgaccc cacaatgagt gcggcggcgc catcactgaa aagagcctga ccgacgaggg
180
agtcaaggtg ggtgtcagag ggaccacgga aggtaacggc tgtgatctca gagcagacaa
240
cgaggacacg tgctccacga ttgttctcgg cgagatcctt agcgatacgg aggacagtac
300
<210> 31
<211> 4035
<212> ДНК
<213> Arabidopsis thaliana
<400> 31
atgggatcta gggttccaat agaaaccatc gaagaagacg gcgaattcga ttgggaagca
60
gcagtcaaag aaatcgactt ggcttgtctt aaaaccacaa acgcttcttc ttcttcgtca
120
tcccatttca ctcctttggc taatccacca attacggcaa atctcactaa gccacctgcg
180
aagagacaat ctactctcga taaattcatc ggcagaaccg aacataaacc ggagaatcat
240
caagttgttt ccgagtgtgg tgttaacgat aacgataata gtcctttagt tgggattgat
300
cctgaggcag ctaaaacttg gatttatcca gtgaatggga gtgttccttt aagagattat
360
cagtttgcta taacgaagac tgctttgttt tcgaatacat tggtggcttt gcctacggga
420
cttggtaaaa cgcttatagc tgcggttgtt atgtataatt acttcagatg gtttccacaa
480
ggtaaaatag tatttgcggc gccttctagg cctcttgtga tgcagcagat tgaggcgtgt
540
cataatattg ttggaatacc acaagaatgg acgattgact tgacgggtca gacatgtcct
600
tcgaaaagag cttttttgtg gaaaagcaaa cgggttttct ttgtcactcc acaagtgtta
660
gagaaggata tacagtcagg aacatgtctt actaactact tggtttgctt ggtgatcgac
720
gaggcacatc gagctttagg gaattattct tattgtgttg tagttcgtga gttgatggcg
780
gtaccgatac agctgagaat actggctctt actgcaactc ctggatcaaa gacacaggcc
840
atccagggta tcattgataa tttgcagata tccacacttg aatatcgaaa tgagagtgac
900
catgatgttt gcccttatgt ccacgacaga aaattagaag tcatcgaggt tcccttgggt
960
caagatgcag atgatgtatc gaaacgcctg tttcatgtta tacgtccata tgcagtcagg
1020
cttaaaaact ttggggttaa tctaaataga gatatacaaa ctttaagtcc acacgaagta
1080
cttatggcaa gggataagtt tcgtcaagca cctctaccag gccttcccca tgtaaatcac
1140
ggagatgtag aatcttgctt tgcagctctt atcactcttt atcatattcg taagctcctt
1200
tctagtcatg gaataagacc agcgtatgag atgctagaag agaaattgaa agaagggcca
1260
tttgctaggt tgatgagtaa gaatgaagat attaggatga cgaagctttt gatgcagcaa
1320
aggttgtcac atggagcacc aagcccaaaa ttgtcgaaga tgttagaaat actggttgat
1380
catttcaaag tgaaagatcc gaagacatca cgggtcatta ttttctcaaa tttcagagga
1440
agcgtaagag acataatgaa cgcattaagt aatattggag atatggtcaa agcaactgag
1500
tttattggtc aaagttcagg taagacattg aaaggccagt cgcaaaaaat tcagcaggct
1560
gttttggaga aatttagagc tggggggttc aatgttattg tcgcaacatc tattggtgaa
1620
gaaggcttgg atatcatgga agttgaccta gttatatgtt ttgatgctaa tgtatctcct
1680
ctgaggatga ttcaacggat gggaagaact ggaaggaaaa ataatggtcg agttgtagtt
1740
cttgcttgtg aaggatcaga aaagaacagc tatatgcgaa agcaagcaag tggacgggct
1800
attaaaaaac acatgcggaa tggaggaaca aatagtttta attttcatcc tagtccaagg
1860
atgattcccc atgtttataa gccagaagtt cagcatgttg agttttcaat caagcaattc
1920
gttccacgtg gaaagaaact acaagaggag tatgccactg agactccagc tttccagaaa
1980
aagcttacac ctgcagagac gcatatgctc gctaagtatt acaacaaccc cgatgaggaa
2040
aagttgagag tgtccttaat tgcgttccct cacttccaga cattgccatc caaggtgcac
2100
aaagtaatgc attcacgtca aacaggcatg ttaattgacg ctatgcagca cttgcaagag
2160
ccaacttttt cagaacagag taaaagcttc ttcactgagt ttcgagctcc tttgggtgaa
2220
agagaagagc ttgatacagg tctgagggtt actaatgatc caaaagatct acactctgtc
2280
cgtgatttgg aagtcaacac atcacagaga aaggcaaaac aagttgaatc tcccacaagc
2340
accttagaga caacagagaa ggattacgaa gaatcttcac ccacacaccg ttatcttttc
2400
agttcagaat gtgcatccgt tgatactctg gggaacgtct tcgtaatgcc agttcctctt
2460
ttattctttc ctaatgttct ggagtcagac aatacgcctc tgcctaaaac agaaaaacaa
2520
cattcttgcc ggaatacatc tcacattgac ttagttccag tagatacttc ggaaaaacat
2580
cggcaagata atatctcatg caagttaaag gaaagattct cgccagacgg tgccagcgag
2640
acactagaga ctcatagcct tgtgaaaagg aactccacca gagtaggtga agatgatgta
2700
gcgaattctg ttggagaaat tgtgttatca tcggatgaag atgactgtga gggattggag
2760
cttagtccac ggctcactaa cttcatcaag agcggcattg ttccagagtc acctgtctat
2820
gaccaagggg aagcgaacag agaagaagat cttgaatttc ctcagctttc ttcacccatg
2880
aggttcagta acgaattggc aggagagtct tctttccctg agagaaaggt tcagcataag
2940
tgcaacgatt ataacattgt gtctacaacc actgaattga gaactcctca gaaggaggta
3000
ggtttggcca acggaacaga atgcttggct gtttctccta ttcctgagga ttggagaact
3060
cccttggcga atctgacaaa cacaaacagc agcgctcgca aagattggcg ggtgagttct
3120
ggagaaaagt tagaaactct tcgacagcct cgcaagttga agagactacg tagacttgga
3180
gattgctcga gtgctgtaaa ggagaattat cctggtatta cagaggcaga ccatatcaga
3240
tctcgttctc gcggtaaaaa gcacattaga ggtaagaaga agatgatcat ggatgatgat
3300
gtccaagtct tcattgacga ggaagctgag gtctcttcgg gagcagagat gtcggctgat
3360
gagaacgaag atgtgactgg cgattcattt gaagatagtt tcatagatga cggaacaatg
3420
cctacagcaa atactcaagc cgagtctggt aaagttgaca tgatggctgt ttacaggcgt
3480
tctcttctca gccagtcacc attaccggcg agatttcgtg atttagccgc atcaagtctg
3540
agtccttatt ctgctggacc cttgacgaga ataaatgaga gcagaagcga ctcagataaa
3600
tcattgtctt ctcttcgaac accaaaaaca acaaactctg agtcaaacca agatgcaatg
3660
atgataggaa acctttcggt agtacaaatc tcgtcagata gccggaaaag gaaatttagc
3720
ttatgcaact cggcgaatgc ccccgtgatt aacttagaaa gcaagtttgc agctcatgca
3780
caagccacgg agaaggaaag ccatgaaggc gtgagaagca atgcaggtgc gttagagtac
3840
aatgatgatg atgatgatgc attctttgcg acactagact ttgatgcaat ggaagcacaa
3900
gccacattgt tattgtcgaa acagagatcc gaagcaaaag agaaagaaga cgcaacggtt
3960
atacctaatc caggcatgca gagaagtgat ggtatggaga aagatgcacc atcttttgat
4020
cttggtctgt ggtga
4035
<210> 32
<211> 2310
<212> ДНК
<213> Brassica napus
<400> 32
atgtcaaatg aaaataaaaa tataaaaact aaatttcatc ctagttcaag gatgattccc
60
catgtttata agccagaagt tcagcatgtt aagttttcga tcgagcaatt cattccacgt
120
ggaaagaagc tacaagatga gcctgccact gagactccag ctttcaagaa aaagcttaca
180
ccggaagaga tggatatgct cgccaagtat ttcaaaccca acgaggaaaa gtggagagtt
240
tccttgattg ctttccctca cttccaaaca ttgccatcca aagtgcacaa agtaatgcat
300
tcacgccaaa caagcatatt aattgatgct atgcagcatc tgcaagagac aactttgaca
360
gagcaaagta aaagtttctt cattaagtat ggagctcctt tggctgaaag agatgagctt
420
gacgcaggtc tgagggttgg tgatgatccg aaagatttac cctcttccga tgatttggat
480
gtcaacacat cacagagaaa ggcaaaacaa attttagaat ctcccacaag cacattagag
540
actacagaga aggatttcga agcatcttca cccacacact gttatctttt cagttcagaa
600
tgtgcgtccg ttgatactct ggggaaggtc tttgtattgc cggttcctct ctcattctct
660
tctaatgtac cagggtcaga ctgcgtggga agagaaaaag aactttcttc cccgaataag
720
tcccacactg acgttgttcc gatagatagt tcctcaaaac atcggcaaga taatatttca
780
tgcaagttaa agcaaggatt cttgccagat tgtgccaacg agactttgga gtcccaaagc
840
cttttgaaaa ggcactccac cgatgtaggt aaaggagata tagagaattg tgctggagaa
900
attatgatat catcggatga agaagacgac tgtgaggatt tggagcttag tccaaggctc
960
actaacttca tcaagagtgg cgttgttcca gattcacctg tctatgacca agttgcatac
1020
gaagcaaaca gagaagaaga ccttgatctt ccacacacga gtttaactaa tgaattggca
1080
gaagagccat cgacacctga gaaaaaggtt cacattgctt ctacggccaa tgaattcaga
1140
actcctcaga aggaagaaga tttagccaac gaaacagaaa gcttcgctgt ttctccaatg
1200
cctgaggagt ggagaactcc cttggcgaat atcaccaacg caagcagcag cgctagcaaa
1260
gattggcgcg tgagttcggg agaaaagtca gaaactcttc gacagcctcg caagttgaag
1320
agacttcgta gacttggaga ttgctcgagt gctgtgaagg agaataatcc tggtattgca
1380
aagacagacc atatcagatc tcgttctcgc agtgtaaaga acataagagg taaaatgatt
1440
ctgtatttcc ttttgctctg tgttcaaggc aagaagaaga tacgcgcgga taataatgct
1500
agaatcttca ttgaagcgga agctgaggtg tcttcggaat cagaaatgtc ggttgatgag
1560
aacgtagatt tgaccagcga ttcatttgaa gatagcttca tagatgacgg tacaatgcct
1620
acagcaaata ctcaagccga gtgtgctaaa gttgacatga tggccgttta caggtatata
1680
tcgaatcaaa acaagtcttt cttctactat gatttactaa gaatcataag ctatggtttc
1740
cacagacgtt ctctactcag ccaatcacca ttaccggcaa gatttcgtga tgtagctgca
1800
tcaagtccga gtccttattc ttctggtctc ttgaagacaa taaatgagag cagaagcgac
1860
tcagataaat cattgtcttc tcttagaacc ccacaaacaa cgaacaacga gtcaaacaag
1920
gatgcaatgg ccacaggaga cctttcggta gcacaaatct caacagacag ccggaaaagg
1980
aaattcagct tatgcaactc agcgaatgtc ccagtgatta acttggaaaa caagtttgaa
2040
gctcatgcac aagccacgga gaaggaaagc catgaaggtc cgagaagcaa tgcaggtgca
2100
tcacagtaca aggatgagga tgaagatgat gatgcattct acgcgacact ggactttgat
2160
gccatggaag cgcatgcgac attgctattg tcgaaacaaa ggtcagaaac gaaaacaaaa
2220
gaagatgcat cggtgaaacc tcatttgggc aatcagagga atgatggttt gccgaaggat
2280
gggccatctt ttgatcttgg tttgtggtga
2310
<210> 33
<211> 1822
<212> ДНК
<213> Искусственная последовательность
<220>
<223> hpFANCM-At[wt]
<400> 33
ggctcgagaa ccgaattcta atacgactca ctatagggtc aggaacatgt cttactaact
60
acttggtttg cttggtgatc gacgaggcac atcgagcttt agggaattat tcttattgtg
120
ttgtagttcg tgagttgatg gcggtaccga tacagctgag aatactggct cttactgcaa
180
ctcctggatc aaagacacag gccatccagg gtatcattga taatttgcag atatccacac
240
ttgaatatcg aaatgagagt gaccatgatg tttgccctta tgtccccgac agaaaattag
300
aagtcatcga ggttcccttg ggtcaagatg cagatgatgt atcgaaacgc ctgtttcatg
360
ttatacgtcc atatgcagtc aggcttaaaa actttggggt taatctaaat agagatatac
420
aaactttaag tccacacgaa gtacttatgg caagggataa gtttcgtcaa gcacctctac
480
caggccttcc ccatgtaaat cacggagatg tagaatcttg ctttgcagct cttatcaggt
540
aaggaaataa ttattttctt ttttcctttt agtataaaat agttaagtga tgttaattag
600
tatgattata ataatatagt tgttataatt gtgaaaaaat aatttataaa tatattgttt
660
acataaacaa catagtaatg taaaaaaata tgacaagtga tgtgtaagac gaagaagata
720
aaagttgaga gtaagtatat tatttttaat gaatttgatc gaacatgtaa gatgatatac
780
tagcattaat atttgtttta atcataatag taattctagc tggtttgatg aattaaatat
840
caatgataaa atactatagt aaaaataaga ataaataaat taaaataata tttttttatg
900
attaatagtt tattatataa ttaaatatct ataccattac taaatatttt agtttaaaag
960
ttaataaata ttttgttaga aattccaatc tgcttgtaat ttatcaataa acaaaatatt
1020
aaataacaag ctaaagtaac aaataatatc aaactaatag aaacagtaat ctaatgtaac
1080
aaaacataat ctaatgctaa tataacaaag cgcaagatct atcattttat atagtattat
1140
tttcaatcaa cattcttatt aatttctaaa taatacttgt agttttatta acttctaaat
1200
ggattgacta ttaattaaat gaattagtcg aacatgaata aacaaggtaa catgatagat
1260
catgtcattg tgttatcatt gatcttacat ttggattgat tacagttgat aagagctgca
1320
aagcaagatt ctacatctcc gtgatttaca tggggaaggc ctggtagagg tgcttgacga
1380
aacttatccc ttgccataag tacttcgtgt ggacttaaag tttgtatatc tctatttaga
1440
ttaaccccaa agtttttaag cctgactgca tatggacgta taacatgaaa caggcgtttc
1500
gatacatcat ctgcatcttg acccaaggga acctcgatga cttctaattt tctgtcgggg
1560
acataagggc aaacatcatg gtcactctca tttcgatatt caagtgtgga tatctgcaaa
1620
ttatcaatga taccctggat ggcctgtgtc tttgatccag gagttgcagt aagagccagt
1680
attctcagct gtatcggtac cgccatcaac tcacgaacta caacacaata agaataattc
1740
cctaaagctc gatgtgcctc gtcgatcacc aagcaaacca agtagttagt aagacatgtt
1800
cctgaccccg ggatccaagc tt
1822
<210> 34
<211> 1822
<212> ДНК
<213> Искусственная последовательность
<220>
<223> hpFANCM-At[G:U]
<400> 34
ggctcgagaa ccgaattcta atacgactca ctatagggtt aggaatatgt tttattaatt
60
atttggtttg tttggtgatt gatgaggtat attgagtttt agggaattat ttttattgtg
120
ttgtagtttg tgagttgatg gtggtattga tatagttgag aatattggtt tttattgtaa
180
tttttggatt aaagatatag gttatttagg gtattattga taatttgtag atatttatat
240
ttgaatattg aaatgagagt gattatgatg tttgttttta tgtttttgat agaaaattag
300
aagttattga ggtttttttg ggttaagatg tagatgatgt attgaaatgt ttgttttatg
360
ttatatgttt atatgtagtt aggtttaaaa attttggggt taatttaaat agagatatat
420
aaattttaag tttatatgaa gtatttatgg taagggataa gttttgttaa gtatttttat
480
taggtttttt ttatgtaaat tatggagatg tagaattttg ttttgtagtt tttattaggt
540
aaggaaataa ttattttctt ttttcctttt agtataaaat agttaagtga tgttaattag
600
tatgattata ataatatagt tgttataatt gtgaaaaaat aatttataaa tatattgttt
660
acataaacaa catagtaatg taaaaaaata tgacaagtga tgtgtaagac gaagaagata
720
aaagttgaga gtaagtatat tatttttaat gaatttgatc gaacatgtaa gatgatatac
780
tagcattaat atttgtttta atcataatag taattctagc tggtttgatg aattaaatat
840
caatgataaa atactatagt aaaaataaga ataaataaat taaaataata tttttttatg
900
attaatagtt tattatataa ttaaatatct ataccattac taaatatttt agtttaaaag
960
ttaataaata ttttgttaga aattccaatc tgcttgtaat ttatcaataa acaaaatatt
1020
aaataacaag ctaaagtaac aaataatatc aaactaatag aaacagtaat ctaatgtaac
1080
aaaacataat ctaatgctaa tataacaaag cgcaagatct atcattttat atagtattat
1140
tttcaatcaa cattcttatt aatttctaaa taatacttgt agttttatta acttctaaat
1200
ggattgacta ttaattaaat gaattagtcg aacatgaata aacaaggtaa catgatagat
1260
catgtcattg tgttatcatt gatcttacat ttggattgat tacagttgat aagagctgca
1320
aagcaagatt ctacatctcc gtgatttaca tggggaaggc ctggtagagg tgcttgacga
1380
aacttatccc ttgccataag tacttcgtgt ggacttaaag tttgtatatc tctatttaga
1440
ttaaccccaa agtttttaag cctgactgca tatggacgta taacatgaaa caggcgtttc
1500
gatacatcat ctgcatcttg acccaaggga acctcgatga cttctaattt tctgtcgggg
1560
acataagggc aaacatcatg gtcactctca tttcgatatt caagtgtgga tatctgcaaa
1620
ttatcaatga taccctggat ggcctgtgtc tttgatccag gagttgcagt aagagccagt
1680
attctcagct gtatcggtac cgccatcaac tcacgaacta caacacaata agaataattc
1740
cctaaagctc gatgtgcctc gtcgatcacc aagcaaacca agtagttagt aagacatgtt
1800
cctgaccccg ggatccaagc tt
1822
<210> 35
<211> 1818
<212> ДНК
<213> Искусственная последовательность
<220>
<223> hpFANCM-Bn[wt]
<400> 35
ggatccttgg tacctaatac gactcactat agggagaaat tatgatatca tcggatgaag
60
aagacgactg tgaggatttg gagcttagtc caaggctcac taacttcatc aagagtggcg
120
ttgttccaga ttcacctgtc tatgaccaag ttgcatacga agcaaacaga gaagaagacc
180
ttgatcttcc acacacgagt ttaactaatg aattggcaga agagccatcg acacctgaga
240
aaaaggttca cattgcttct acggccaatg aattcagaac cccaacgaag gaagaagatt
300
tagccaacga aacagaaagc ttcgctgttt ctccaatgcc tgaggagtgg agaactccct
360
tggcgaatat caccaacgca agcagcagcg ctagcaaaga ttggcgcgtg agttcgggag
420
aaaagtcaga aactcttcga cagcctcgca agttgaagag acttcgtaga cttggagatt
480
gctcgagtgc tgtgaaggag aataatcctg gtattgcaaa gacagaccat atcgtaagga
540
aataattatt ttcttttttc cttttagtat aaaatagtta agtgatgtta attagtatga
600
ttataataat atagttgtta taattgtgaa aaaataattt ataaatatat tgtttacata
660
aacaacatag taatgtaaaa aaatatgaca agtgatgtgt aagacgaaga agataaaagt
720
tgagagtaag tatattattt ttaatgaatt tgatcgaaca tgtaagatga tatactagca
780
ttaatatttg ttttaatcat aatagtaatt ctagctggtt tgatgaatta aatatcaatg
840
ataaaatact atagtaaaaa taagaataaa taaattaaaa taatattttt ttatgattaa
900
tagtttatta tataattaaa tatctatacc attactaaat attttagttt aaaagttaat
960
aaatattttg ttagaaattc caatctgctt gtaatttatc aataaacaaa atattaaata
1020
acaagctaaa gtaacaaata atatcaaact aatagaaaca gtaatctaat gtaacaaaac
1080
ataatctaat gctaatataa caaagcgcaa gatctatcat tttatatagt attattttca
1140
atcaacattc ttattaattt ctaaataata cttgtagttt tattaacttc taaatggatt
1200
gactattaat taaatgaatt agtcgaacat gaataaacaa ggtaacatga tagatcatgt
1260
cattgtgtta tcattgatct tacatttgga ttgattacag gatatggtct gtctttgcaa
1320
taccaggatt attctccttc acagcactcg agcaatctcc aagtctacga agtctcttca
1380
acttgcgagg ctgtcgaaga gtttctgact tttctcccga actcacgcgc caatctttgc
1440
tagcgctgct gcttgcgttg gtgatattcg ccaagggagt tctccactcc tcaggcattg
1500
gagaaacagc gaagctttct gtttcgttgg ctaaatcttc ttccttcgtt ggggttctga
1560
attcattggc cgtagaagca atgtgaacct ttttctcagg tgtcgatggc tcttctgcca
1620
attcattagt taaactcgtg tgtggaagat caaggtcttc ttctctgttt gcttcgtatg
1680
caacttggtc atagacaggt gaatctggaa caacgccact cttgatgaag ttagtgagcc
1740
ttggactaag ctccaaatcc tcacagtcgt cttcttcatc cgatgatatc ataatttctc
1800
tctagaaagg atcccggg
1818
<210> 36
<211> 1818
<212> ДНК
<213> Искусственная последовательность
<220>
<223> hpFANCM-Bn[G:U]
<400> 36
ggatccttgg tacctaatac gactcactat agggagaaat tatgatatta ttggatgaag
60
aagatgattg tgtggatttg gagtttagtt taaggtttat taattttatt aagagtggtg
120
ttgttttaga tttatttgtt tatgattaag ttgtatatga agtaaatagt gaagaagatt
180
ttgatttttt atatatgagt ttaattaatg aattggtaga agagttattg atatttgaga
240
aaaaggttta tattgttttt atggttaatg aatttagaat tttaatgaag gaagaagatt
300
tagttaatga aatagaaagt tttgttgttt ttttaatgtt tgaggagtgg agaatttttt
360
tggtgaatat tattaatgta agtagtagtg ttagtaaaga ttggtgtgtg agtttgggag
420
aaaagttaga aattttttga tagttttgta agttgaagag attttgtaga tttggagatt
480
gtttgagtgt tgtgaaggag aataattttg gtattgtaaa gatagattat attgtaagga
540
aataattatt ttcttttttc cttttagtat aaaatagtta agtgatgtta attagtatga
600
ttataataat atagttgtta taattgtgaa aaaataattt ataaatatat tgtttacata
660
aacaacatag taatgtaaaa aaatatgaca agtgatgtgt aagacgaaga agataaaagt
720
tgagagtaag tatattattt ttaatgaatt tgatcgaaca tgtaagatga tatactagca
780
ttaatatttg ttttaatcat aatagtaatt ctagctggtt tgatgaatta aatatcaatg
840
ataaaatact atagtaaaaa taagaataaa taaattaaaa taatattttt ttatgattaa
900
tagtttatta tataattaaa tatctatacc attactaaat attttagttt aaaagttaat
960
aaatattttg ttagaaattc caatctgctt gtaatttatc aataaacaaa atattaaata
1020
acaagctaaa gtaacaaata atatcaaact aatagaaaca gtaatctaat gtaacaaaac
1080
ataatctaat gctaatataa caaagcgcaa gatctatcat tttatatagt attattttca
1140
atcaacattc ttattaattt ctaaataata cttgtagttt tattaacttc taaatggatt
1200
gactattaat taaatgaatt agtcgaacat gaataaacaa ggtaacatga tagatcatgt
1260
cattgtgtta tcattgatct tacatttgga ttgattacag gatatggtct gtctttgcaa
1320
taccaggatt attctccttc acagcactcg agcaatctcc aagtctacga agtctcttca
1380
acttgcgagg ctgtcgaaga gtttctgact tttctcccga actcacgcgc caatctttgc
1440
tagcgctgct gcttgcgttg gtgatattcg ccaagggagt tctccactcc tcaggcattg
1500
gagaaacagc gaagctttct gtttcgttgg ctaaatcttc ttccttcgtt ggggttctga
1560
attcattggc cgtagaagca atgtgaacct ttttctcagg tgtcgatggc tcttctgcca
1620
attcattagt taaactcgtg tgtggaagat caaggtcttc ttctctgttt gcttcgtatg
1680
caacttggtc atagacaggt gaatctggaa caacgccact cttgatgaag ttagtgagcc
1740
ttggactaag ctccaaatcc tcacagtcgt cttcttcatc cgatgatatc ataatttctc
1800
tctagaaagg atcccggg
1818
<210> 37
<211> 2274
<212> ДНК
<213> Brassica napus
<400> 37
atggttagtc tgcgctccac agaaaacact ccggcttcgg aaatggccag cgacggcaaa
60
acggagaaag atggctccgg cgactcaccc acttctgttc tcagcgatga ggaaaactgt
120
gaagagaaaa ctgctactgt tgctgtagag gaagagatac ttctagccaa gaatggagat
180
tcgtctctta tctctgaggc catggctcag gaagaagagc agcttctcaa aatccgggaa
240
gatgaagaga ttgctaaacg tgctgctggc tctggtgaag ctcctgatct gaatgatact
300
cagtttacta aacttgatga gctcttgacc caaacccagc tctactctga gtttctcctt
360
gagaaaatgg aggatatcac caaaaatggg atagaaggtg agacccaaaa ggccgagcct
420
gagcctgagc ctgagcccga gaagaaaggc cgtggacgta aaagaaaggc tgctcctcag
480
ggcgacagta tgaaggctaa gaaagctgtt gctgctatga tttcaagatc caaagaaggc
540
cgtgaatctg ccgactcaga tctgacagag gaagaaagag tcatgaaaga gcagggtgaa
600
cttgttcctc ttctgactgg cggaaagtta aagtcttatc agctcaaagg tgtcaaatgg
660
ctgatatcat tgtggcaaaa tggtttgaat ggaattttag ctgatcaaat gggtcttgga
720
aagacaattc aaaccattgg tttcctatca cacctcaaag gaaatgggtt ggatggtcca
780
tatctagtca ttgccccact ctctactctt tcaaactgga tgaatgagat cgctaggttc
840
acgccttcca ttaatgcaat catttaccat ggagataaga aagaaaggga tgagctcagg
900
aagaggcaca tgcccagaac tgttggtccg aagttcccta tagtcataac ttcttatgag
960
gttgctatga atgatgctaa aaagaatctg cggcactatc catggaaata tgttgtgatt
1020
gatgagggtc acaggttgaa aaaccacaag tgtaaactgc tgagggagct aagatacttg
1080
aatatggaga acaaacttct gctgacagga acacctctgc aaaataattt gtctgagctt
1140
tggtcactgt tgaattttat tctgcctgac atctttgcat cacatgacga atttgaatca
1200
tggtttgatt tttctggaaa gaataataat gaagcaacta aggaagaagg agaagagaaa
1260
agaagagctc aagtggttgc gaaacttcat aatatactac gacctttcat cctccggaga
1320
atgaaatgtg atgttgagct ctcacttccc cggaaaaaag agattatcat ctatgctaca
1380
atgacggacc atcagaagaa gttccaggaa catcttgtga accacacctt ggaagcacac
1440
attagagatg atactgtccg aggtcatggc ttgaagggaa agcttaacaa tcttgctatt
1500
caacttcgaa agaactgcaa ccatcctgac cttcttgtgg ggcaactaga tggctcatat
1560
ctctacccac ctttggaaga cattgtggga cagtgcggta aattccgctt attggagaga
1620
ttgcttgttc ggttatttgc caaaaatcac agagtcctta tcttctccca gtggacaaaa
1680
atactggaca ttatggatta ctacttcagt gagaaggggt ttgaggtttg ccgaatcgac
1740
ggtagtgtga aactagaaga aaggagaaga cagatccaag aattcaatga tgagaagagc
1800
aactgcagga tatttcttct cagtaccaga gccggaggac tcggaattaa tcttactgct
1860
gcagatacat gcatcctcta cgatagcgat tggaaccctc aaatggactt gcaagccatg
1920
gacagatgcc acagaattgg tcagacaaaa cctgttcatg tttacaggct tgcgacggct
1980
cagtcaatag agggccgagt tctgaaacga gcatacagta agcttaagct ggaacatgtg
2040
gttattggca aggggcagtt tcatcaagaa cgtgccaagt cttcaacacc gttagaggaa
2100
gatgacatac tggcgttgct taaggacgac gaaaatgctg aagataaact gatacaaacc
2160
gacataagcg aggaggatct tgacagggtg cttgaccgta gtgatctgat gattacctta
2220
ccgggcgaga ctcaagcaca tgaagctttt ccagtgaagg gtccgggttg ggaa
2274
<210> 38
<211> 1824
<212> ДНК
<213> Искусственная последовательность
<220>
<223> hpDDM1-Bn[wt]
<400> 38
ggatccttgg tacctaatac gactcactat agggagctgt tgctgctatg atttcaagat
60
ccaaagaagg ccgtgaatct gccgactcag atctgacaga ggaagaaaga gtcatgaatg
120
agcagggtga acttgttcct cttctgactg gcggaaagtt aaagtcttat cagctcaaag
180
gtgtcaaatg gctgatatca ttgtggcaaa atggtttgaa tggaatttta gctgatcaaa
240
tgggtcttgg aaagacaatt caaaccattg gtttcctatc accccaacaa ggaaatgggt
300
tggatggtcc atatctagtc attgccccac tctctactct ttcaaaagcg attggaaccc
360
tcaaatggac ttgcaagcca tggacagatg ccacagaatt ggtcagacaa aacctgttca
420
tgtttacagg cttgcgacgg ctcagtcaat agagggccga gttctgaaac gagcatacag
480
taagcttaag ctggaacatg tggttattgg caaggggcag tttcatcaag aacgtggtaa
540
ggaaataatt attttctttt ttccttttag tataaaatag ttaagtgatg ttaattagta
600
tgattataat aatatagttg ttataattgt gaaaaaataa tttataaata tattgtttac
660
ataaacaaca tagtaatgta aaaaaatatg acaagtgatg tgtaagacga agaagataaa
720
agttgagagt aagtatatta tttttaatga atttgatcga acatgtaaga tgatatacta
780
gcattaatat ttgttttaat cataatagta attctagctg gtttgatgaa ttaaatatca
840
atgataaaat actatagtaa aaataagaat aaataaatta aaataatatt tttttatgat
900
taatagttta ttatataatt aaatatctat accattacta aatattttag tttaaaagtt
960
aataaatatt ttgttagaaa ttccaatctg cttgtaattt atcaataaac aaaatattaa
1020
ataacaagct aaagtaacaa ataatatcaa actaatagaa acagtaatct aatgtaacaa
1080
aacataatct aatgctaata taacaaagcg caagatctat cattttatat agtattattt
1140
tcaatcaaca ttcttattaa tttctaaata atacttgtag ttttattaac ttctaaatgg
1200
attgactatt aattaaatga attagtcgaa catgaataaa caaggtaaca tgatagatca
1260
tgtcattgtg ttatcattga tcttacattt ggattgatta cagtcacgtt cttgatgaaa
1320
ctgccccttg ccaataacca catgttccag cttaagctta ctgtatgctc gtttcagaac
1380
tcggccctct attgactgag ccgtcgcaag cctgtaaaca tgaacaggtt ttgtctgacc
1440
aattctgtgg catctgtcca tggcttgcaa gtccatttga gggttccaat cgcttttgaa
1500
agagtagaga gtggggcaat gactagatat ggaccatcca acccatttcc ttgttggggt
1560
gataggaaac caatggtttg aattgtcttt ccaagaccca tttgatcagc taaaattcca
1620
ttcaaaccat tttgccacaa tgatatcagc catttgacac ctttgagctg ataagacttt
1680
aactttccgc cagtcagaag aggaacaagt tcaccctgct ctttcatgac tctttcttcc
1740
tctgtcagat ctgagtcggc agattcacgg ccttctttgg atcttgaaat catagcagca
1800
acagcttcta gaaaggatcc cggg
1824
<210> 39
<211> 1823
<212> ДНК
<213> Искусственная последовательность
<220>
<223> hpDDM1-Bn[G:U]
<400> 39
ggatccttgg tacctaatac gactcactat agggttgttg ttgttatgat tttaagattt
60
aaagaaggtt gtgaatttgt tgatttagat ttgatagagg aagaaagagt tatgaatgag
120
tagggtgaat ttgttttttt tttgattggt ggaaagttaa agttttatta gtttaaaggt
180
gttaaatggt tgatattatt gtggtaaaat ggtttgaatg gaattttagt tgattaaatg
240
ggttttggaa agataattta aattattggt tttttattat attttaaagg aaatgggttg
300
gatggtttat atttagttat tgttttattt tttatttttt taaaagtgat tggaattttt
360
aaatggattt gtaagttatg gatagatgtt atagaattgg ttagataaaa tttgtttatg
420
tttataggtt tgtgatggtt tagttaatag agggttgagt tttgaaatga gtatatagta
480
agtttaagtt ggaatatgtg gttattggta aggggtagtt ttattaagaa tgtgtggtaa
540
ggaaataatt attttctttt ttccttttag tataaaatag ttaagtgatg ttaattagta
600
tgattataat aatatagttg ttataattgt gaaaaaataa tttataaata tattgtttac
660
ataaacaaca tagtaatgta aaaaaatatg acaagtgatg tgtaagacga agaagataaa
720
agttgagagt aagtatatta tttttaatga atttgatcga acatgtaaga tgatatacta
780
gcattaatat ttgttttaat cataatagta attctagctg gtttgatgaa ttaaatatca
840
atgataaaat actatagtaa aaataagaat aaataaatta aaataatatt tttttatgat
900
taatagttta ttatataatt aaatatctat accattacta aatattttag tttaaaagtt
960
aataaatatt ttgttagaaa ttccaatctg cttgtaattt atcaataaac aaaatattaa
1020
ataacaagct aaagtaacaa ataatatcaa actaatagaa acagtaatct aatgtaacaa
1080
aacataatct aatgctaata taacaaagcg caagatctat cattttatat agtattattt
1140
tcaatcaaca ttcttattaa tttctaaata atacttgtag ttttattaac ttctaaatgg
1200
attgactatt aattaaatga attagtcgaa catgaataaa caaggtaaca tgatagatca
1260
tgtcattgtg ttatcattga tcttacattt ggattgatta cagtcacgtt cttgatgaaa
1320
ctgccccttg ccaataacca catgttccag cttaagctta ctgtatgctc gtttcagaac
1380
tcggccctct attgactgag ccgtcgcaag cctgtaaaca tgaacaggtt ttgtctgacc
1440
aattctgtgg catctgtcca tggcttgcaa gtccatttga gggttccaat cgcttttgaa
1500
agagtagaga gtggggcaat gactagatat ggaccatcca acccatttcc ttgttggggt
1560
gataggaaac caatggtttg aattgtcttt ccaagaccca tttgatcagc taaaattcca
1620
ttcaaaccat tttgccacaa tgatatcagc catttgacac ctttgagctg ataagacttt
1680
aactttccgc cagtcagaag aggaacaagt tcaccctgct ctttcatgac tctttcttcc
1740
tctgtcagat ctgagtcggc agattcacgg ccttctttgg atcttgaaat catagcagca
1800
acagttctag aaaggatccc ggg
1823
<210> 40
<211> 720
<212> ДНК
<213> Искусственная последовательность
<220>
<223> EGFP
<400> 40
atggtgagca agggcgagga gctgttcacc ggggtggtgc ccatcctggt cgagctggac
60
ggcgacgtaa acggccacaa gttcagcgtg tccggcgagg gcgagggcga tgccacctac
120
ggcaagctga ccctgaagtt catctgcacc accggcaagc tgcccgtgcc ctggcccacc
180
ctcgtgacca ccttcaccta cggcgtgcag tgcttcagcc gctaccccga ccacatgaag
240
cagcacgact tcttcaagtc cgccatgccc gaaggctacg tccaggagcg caccatcttc
300
ttcaaggacg acggcaacta caagacccgc gccgaggtga agttcgaggg cgacaccctg
360
gtgaaccgca tcgagctgaa gggcatcgac ttcaaggagg acggcaacat cctggggcac
420
aagctggagt acaactacaa cagccacaac gtctatatca tggccgacaa gcagaagaac
480
ggcatcaagg tgaacttcaa gatccgccac aacatcgagg acggcagcgt gcagctcgcc
540
gaccactacc agcagaacac ccccatcggc gacggccccg tgctgctgcc cgacaaccac
600
tacctgagca cccagtccgc cctgagcaaa gaccccaacg agaagcgcga tcacatggtc
660
ctgctggagt tcgtgaccgc cgccgggatc actctcggca tggacgagct gtacaagtaa
720
<210> 41
<211> 1262
<212> ДНК
<213> Искусственная последовательность
<220>
<223> hpEGFP[wt]
<400> 41
gctagctaat acgactcact atagggcagc agcacggggc cgtcgccgat gggggtgttc
60
tgctggtagt ggtcggcgag ctgcacgctg ccgtcctcga tgttgtggcg gatcttgaag
120
ttcaccttga tgccgttctt ctgcttgtcg gccatgatgt atacgttgtg gctgttgaag
180
ttgtactcca gcttgtgccc caggatgttg ccgtcctcct tgaagtcgat gcccttcagc
240
tcgatgcggt tcaccagggt gtcgccctcg aacttcacct cggcgcgggt cttgtagttg
300
ccgtcgtcct tgaagaagat ggtgcgctcc tggacgtagc cttcgggcat ggcggacttg
360
aagaagtcgt gctgcttcat gtggtcgggg tagcggctga agcactgcac gccgtaagcg
420
aaggtggtca ctagtgtggg ccagggcacg ggcagcttgc cggtggtgca gatgaacttc
480
agggtctaga ccgcgtcggc atccggtcag tggcagtgaa gggcgaacag ttcctgatta
540
gggggatgaa gctacctggt ccgaaccaca aaccgttcta ctttactggc tttggtcgtc
600
atgaagatgc ggacttgcgt ggcaaaggat tcgataacgt gctgatggtg cacgaccacg
660
cattaatgga ctttaccttt taatggggaa tgaagctacc tggtccgaac tcctaccgta
720
cctcgcatta cccttacgct gaagagatgc tcgactgggc agatgaacat ggcatcgtat
780
ttaggtgaca ctatagccct gaagttcatc tgcaccaccg gcaagctgcc cgtgccctgg
840
cccacactag tgaccacctt cgcttacggc gtgcagtgct tcagccgcta ccccgaccac
900
atgaagcagc acgacttctt caagtccgcc atgcccgaag gctacgtcca ggagcgcacc
960
atcttcttca aggacgacgg caactacaag acccgcgccg aggtgaagtt cgagggcgac
1020
accctggtga accgcatcga gctgaagggc atcgacttca aggaggacgg caacatcctg
1080
gggcacaagc tggagtacaa cttcaacagc cacaacgtat acatcatggc cgacaagcag
1140
aagaacggca tcaaggtgaa cttcaagatc cgccacaaca tcgaggacgg cagcgtgcag
1200
ctcgccgacc actaccagca gaacaccccc atcggcgacg gccccgtgct gctgccgtcg
1260
ac
1262
<210> 42
<211> 1262
<212> ДНК
<213> Искусственная последовательность
<220>
<223> hpEGFP[G:U]
<400> 42
gctagctaat acgactcact atagggcagc agcacggggc cgtcgccgat gggggtgttc
60
tgctggtagt ggtcggcgag ctgcacgctg ccgtcctcga tgttgtggcg gatcttgaag
120
ttcaccttga tgccgttctt ctgcttgtcg gccatgatgt atacgttgtg gctgttgaag
180
ttgtactcca gcttgtgccc caggatgttg ccgtcctcct tgaagtcgat gcccttcagc
240
tcgatgcggt tcaccagggt gtcgccctcg aacttcacct cggcgcgggt cttgtagttg
300
ccgtcgtcct tgaagaagat ggtgcgctcc tggacgtagc cttcgggcat ggcggacttg
360
aagaagtcgt gctgcttcat gtggtcgggg tagcggctga agcactgcac gccgtaagcg
420
aaggtggtca ctagtgtggg ccagggcacg ggcagcttgc cggtggtgca gatgaacttc
480
agggtctaga ccgcgtcggc atccggtcag tggcagtgaa gggcgaacag ttcctgatta
540
gggggatgaa gctacctggt ccgaaccaca aaccgttcta ctttactggc tttggtcgtc
600
atgaagatgc ggacttgcgt ggcaaaggat tcgataacgt gctgatggtg cacgaccacg
660
cattaatgga ctttaccttt taatggggaa tgaagctacc tggtccgaac tcctaccgta
720
cctcgcatta cccttacgct gaagagatgc tcgactgggc agatgaacat ggcatcgtat
780
ttaggtgaca ctatagcctt gaagtttatt tgtattattg gtaagttgtt tgtgttttgg
840
tttatattag tgattatttt tgtttatggt gtgtagtgtt ttagttgtta ttttgattat
900
atgaagtagt atgatttttt taagtttgtt atgtttgaag gttatgttta ggagtgtatt
960
atttttttta aggatgatgg taattataag atttgtgttg aggtgaagtt tgagggtgat
1020
attttggtga attgtattga gttgaagggt attgatttta aggaggatgg taatattttg
1080
gggtataagt tggagtataa ttttaatagt tataatgtat atattatggt tgataagtag
1140
aagaatggta ttaaggtgaa ttttaagatt tgttataata ttgaggatgg tagtgtgtag
1200
tttgttgatt attattagta gaatattttt attggtgatg gttttgtgtt gttgttgtcg
1260
ac
1262
<210> 43
<211> 1259
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ledEGFP[wt]
<400> 43
gctagctaat acgactcact atagggtgtc gccctcgaac ttcacctcgg cgcgggtctt
60
gtagttgccg tcgtccttga agaagatggt gcgctcctgg acgtagcctt cgggcatggc
120
ggacttgaag aagtcgtgct gcttcatgtg gtcggggtag cggctgaagc actgcacgcc
180
gtaggtgaag gtggtcacga gggtgggcca gggcacgggc agcttgccgg tggtgcagat
240
gaacttcagg gtctagaccg cgtcggcatc cggtcagtgg cagtgaaggg cgaacagttc
300
ctgattaggg ggatgaagct acctggtccg aaccacaaac cgttctactt tactggcttt
360
ggtcgtcatg aagatgcgga cttgcgtggc aaaggattcg accctgaagt tcatctgcac
420
caccggcaag ctgcccgtgc cctggcccac cctcgtgacc accttcacct acggcgtgca
480
gtgcttcagc cgctaccccg accacatgaa gcagcacgac ttcttcaagt ccgccatgcc
540
cgaaggctac gtccaggagc gcaccatctt cttcaaggac gacggcaact acaagacccg
600
cgccgaggtg aagttcgagg gcgacaccct ggtgaaccgc atcgagctga agggcatcga
660
cttcaaggag gacggcaaca tcctggggca caagctggag tacaactaca acagccacaa
720
cgtctatatc atggccgaca agcagaagaa cggcatcaag gtgaacttca agatccgcca
780
caacatcgag gacggcagcg tgcagctcgc cgaccactac cagcagaaca cccccatcgg
840
cgacggcccc gtgctgctgc ctaacgtgct gatggtgcac gaccacgcat taatggactt
900
taccttttaa tggggaatga agctacctgg tccgaactcc taccgtacct cgcattaccc
960
ttacgctgaa gagatgctcg actgggcaga tgaacatggc atcgtggcag cagcacgggg
1020
ccgtcgccga tgggggtgtt ctgctggtag tggtcggcga gctgcacgct gccgtcctcg
1080
atgttgtggc ggatcttgaa gttcaccttg atgccgttct tctgcttgtc ggccatgata
1140
tagacgttgt ggctgttgta gttgtactcc agcttgtgcc ccaggatgtt gccgtcctcc
1200
ttgaagtcga tgcccttcag ctcgatgcgg ttcaccattg tcgggataca ctcgtcgac
1259
<210> 44
<211> 1259
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ledEGFP[G:U]
<400> 44
gctagctaat acgactcact atagggtgtc gccctcgaac ttcacctcgg cgcgggtctt
60
gtagttgccg tcgtccttga agaagatggt gcgctcctgg acgtagcctt cgggcatggc
120
ggacttgaag aagtcgtgct gcttcatgtg gtcggggtag cggctgaagc actgcacgcc
180
gtaggtgaag gtggtcacga gggtgggcca gggcacgggc agcttgccgg tggtgcagat
240
gaacttcagg gtctagaccg cgtcggcatc cggtcagtgg cagtgaaggg cgaacagttc
300
ctgattaggg ggatgaagct acctggtccg aaccacaaac cgttctactt tactggcttt
360
ggtcgtcatg aagatgcgga cttgcgtggc aaaggattcg attttgaagt ttatttgtat
420
tattggtaag ttgtttgtgt tttggtttat ttttgtgatt atttttattt atggtgtgta
480
gtgttttagt tgttattttg attatatgaa gtagtatgat ttttttaagt ttgttatgtt
540
tgaaggttat gtttaggagt gtattatttt ttttaaggat gatggtaatt ataagatttg
600
tgttgaggtg aagtttgagg gtgatatttt ggtgaattgt attgagttga agggtattga
660
ttttaaggag gatggtaata ttttggggta taagttggag tataattata atagttataa
720
tgtttatatt atggttgata agtagaagaa tggtattaag gtgaatttta agatttgtta
780
taatattgag gatggtagtg tgtagtttgt tgattattat tagtagaata tttttattgg
840
tgatggtttt gtgttgttgt ttaacgtgct gatggtgcac gaccacgcat taatggactt
900
taccttttaa tggggaatga agctacctgg tccgaactcc taccgtacct cgcattaccc
960
ttacgctgaa gagatgctcg actgggcaga tgaacatggc atcgtggcag cagcacgggg
1020
ccgtcgccga tgggggtgtt ctgctggtag tggtcggcga gctgcacgct gccgtcctcg
1080
atgttgtggc ggatcttgaa gttcaccttg atgccgttct tctgcttgtc ggccatgata
1140
tagacgttgt ggctgttgta gttgtactcc agcttgtgcc ccaggatgtt gccgtcctcc
1200
ttgaagtcga tgcccttcag ctcgatgcgg ttcaccattg tcgggataca ctcgtcgac
1259
<210> 45
<211> 200
<212> ДНК
<213> Искусственная последовательность
<220>
<223> hpGUS[G:U]
<400> 45
ttgtgttggt atttggttag tggtagtgaa gggtgaatag tttttgatta attataaatt
60
gttttatttt attggttttg gttgttatga agatgtggat ttgtgtggta aaggatttga
120
taatgtgttg atggtgtatg attatgtatt aatggattgg attggggtta atttttattg
180
tattttgtat tatttttatg
200
<210> 46
<211> 200
<212> ДНК
<213> Искусственная последовательность
<220>
<223> hpGUS[1:4]
<400> 46
tcgggtccgc aaccgctcac tgggagtcaa gcgcgtacac ttcgtgaata agcactaacg
60
gttgtacatt agtgggtttc gtcctcaaga acatggggag ttgggtgcca atggaatcgt
120
taaggtggtg aaggtccacc acctcgcttt attggtctgc attcggggca agtccaaccc
180
tacgtcggat ttcccatacc
200
<210> 47
<211> 200
<212> ДНК
<213> Искусственная последовательность
<220>
<223> hpGUS[2:10]
<400> 47
tcgcgtcgcg atccggtctc tggcagtgtt gggcgaactc ttcctgatat accacaaagg
60
gttctactaa actggcttac gtcgtcatct agatgcggtg ttgcgtgggt aaggattcct
120
taacgtgcac atggtgcagc accacgcaaa aatggactcc attggggcgt actcctacgc
180
tacctcgcta tacccttagc
200
<210> 48
<211> 200
<212> ДНК
<213> Искусственная последовательность
<220>
<223> hpEIN2[G:U]
<400> 48
gattttagtt agggtttatt tagagaatgg tttttgtttt attttttgtt tttttggttt
60
ttgttggata tattgatttt gggaaatggg ttgtaaatat tgaaggaggt gtttgttttg
120
ggtatgattt ggtggtaatt attttgtttt ttaattttgt tgttatttta tgttaatatg
180
ttgtagtttg tataagtgtt
200
<210> 49
<211> 200
<212> ДНК
<213> Искусственная последовательность
<220>
<223> hpCHS[G:U]
<400> 49
gttgtttgtt ttgagattat agttgttatt ttttgtggtt tttttgatat ttattttgat
60
ttttttgttg gttaggtttt ttttagtgat ggtgttgttg tatttattgt ggggttggat
120
tttgatatat ttgttggaga gaaatttatt tttgagatgg tgtttgttgt ttagattatt
180
tttttagatt ttgatggtgt
200
<210> 50
<211> 200
<212> ДНК
<213> Искусственная последовательность
<220>
<223> hpEIN2[G:U/U:G]
<400> 50
aatgtttatg tgagttgtaa tatattggta taagatggtg gtaaaattga aaagtagagt
60
aattgttatt aagttatatt tgaaatgagt attttttttg atatttgtaa tttatttttt
120
gggattaata tatttgatag aaattaaaag gataggaagt agagtaggaa ttattttttg
180
gataaatttt agttgaggtt
200
<210> 51
<211> 199
<212> ДНК
<213> Искусственная последовательность
<220>
<223> hpCHS[G:U/U:G]
<400> 51
gtattattag agtttggaag gatggtttga gtggtagata ttattttaaa gatgggtttt
60
tttttgatag atgtgttagg gtttgatttt ataatgagtg tggtggtgtt attattgaaa
120
agagtttgat tgatgaggga gttaaggtgg gtgttagagg gattatggaa ggtaatggtt
180
gtgattttag agtagataa
199
<210> 52
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> GUS-WT-F
<400> 52
cctcgaggat cctcgcgtcg gcatccggtc
30
<210> 53
<211> 33
<212> ДНК
<213> Искусственная последовательность
<220>
<223> GUS-WT-R
<400> 53
gggtaccaag cttcgtaagg gtaatgcgag gta
33
<210> 54
<211> 118
<212> ДНК
<213> Искусственная последовательность
<220>
<223> GUS-GU-F
<400> 54
ccctcgagtt gtgttggtat ttggttagtg gtagtgaagg gtgaatagtt tttgattaat
60
tataaattgt tttattttat tggttttggt tgttatgaag atgtggattt gtgtggta
118
<210> 55
<211> 118
<212> ДНК
<213> Искусственная последовательность
<220>
<223> GUS-GU-R
<400> 55
ggggtaccca taaaaataat acaaaataca ataaaaatta accccaatcc aatccattaa
60
tacataatca tacaccatca acacattatc aaatccttta ccacacaaat ccacatct
118
<210> 56
<211> 118
<212> ДНК
<213> Искусственная последовательность
<220>
<223> GUS-4M-F
<400> 56
ccctcgagtc gggtccgcaa ccgctcactg ggagtcaagc gcgtacactt cgtgaataag
60
cactaacggt tgtacattag tgggtttcgt cctcaagaac atggggagtt gggtgcca
118
<210> 57
<211> 118
<212> ДНК
<213> Искусственная последовательность
<220>
<223> GUS-4M-R
<400> 57
ggggtaccgg tatgggaaat ccgacgtagg gttggacttg ccccgaatgc agaccaataa
60
agcgaggtgg tggaccttca ccaccttaac gattccattg gcacccaact ccccatgt
118
<210> 58
<211> 118
<212> ДНК
<213> Искусственная последовательность
<220>
<223> GUS-10M-F
<400> 58
ccctcgagtc gcgtcgcgat ccggtctctg gcagtgttgg gcgaactctt cctgatatac
60
cacaaagggt tctactaaac tggcttacgt cgtcatctag atgcggtgtt gcgtgggt
118
<210> 59
<211> 118
<212> ДНК
<213> Искусственная последовательность
<220>
<223> GUS-10M-R
<400> 59
ggggtaccgc taagggtata gcgaggtagc gtaggagtac gccccaatgg agtccatttt
60
tgcgtggtgc tgcaccatgt gcacgttaag gaatccttac ccacgcaaca ccgcatct
118
<210> 60
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер (35S-F3)
<400> 60
tggctcctac aaatgccatc
20
<210> 61
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Обратный праймер (GUSwt-R2)
<220>
<221> R
<222> (3)..(4)
<223> A или G
<220>
<221> R
<222> (9)..(9)
<223> A или G
<220>
<221> R
<222> (13)..(13)
<223> A или G
<400> 61
carraactrt tcrcccttca c
21
<210> 62
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер (GUSgu-R2)
<400> 62
caaaaactat tcacccttca c
21
<210> 63
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Обратный праймер (GUS4m-R2)
<220>
<221> R
<222> (4)..(4)
<223> A или G
<220>
<221> R
<222> (7)..(7)
<223> A или G
<220>
<221> R
<222> (9)..(9)
<223> A или G
<220>
<221> R
<222> (13)..(13)
<223> A или G
<220>
<221> R
<222> (15)..(15)
<223> A или G
<220>
<221> R
<222> (19)..(19)
<223> A или G
<400> 63
cacraartrt acrcrcttra c
21
<210> 64
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер (35S-F2)
<400> 64
gaggatctaa cagaactcgc
20
<210> 65
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Обратный праймер (35S-R1)
<400> 65
ctctccaaat gaaatgaact tcc
23
<210> 66
<211> 36
<212> ДНК
<213> Искусственная последовательность
<220>
<223> EIN2wt-F
<400> 66
cctcgaggat cctctagacc tcagctaggg tttatc
36
<210> 67
<211> 33
<212> ДНК
<213> Искусственная последовательность
<220>
<223> EIN2wt-R
<400> 67
gggtaccaag cttaacgctt atgcgagctg caa
33
<210> 68
<211> 32
<212> ДНК
<213> Искусственная последовательность
<220>
<223> CHSwt-F
<400> 68
cctcgaggat ccgttgtctg ctctgagatc ac
32
<210> 69
<211> 38
<212> ДНК
<213> Искусственная последовательность
<220>
<223> CHSwt-R
<400> 69
gggtaccaag cttctagagc accatcagag tctggaag
38
<210> 70
<211> 119
<212> ДНК
<213> Искусственная последовательность
<220>
<223> EIN2gu-F
<400> 70
cctcgagtct agattttagt tagggtttat ttagagaatg gtttttgttt tattttttgt
60
ttttttggtt tttgttggat atattgattt tgggaaatgg gttgtaaata ttgaaggag
119
<210> 71
<211> 119
<212> ДНК
<213> Искусственная последовательность
<220>
<223> EIN2gu-R
<400> 71
gggtaccaac acttatacaa actacaacat attaacataa aataacaaca aaattaaaaa
60
acaaaataat taccaccaaa tcatacccaa aacaaacacc tccttcaata tttacaacc
119
<210> 72
<211> 119
<212> ДНК
<213> Искусственная последовательность
<220>
<223> CHSgu-F
<400> 72
cctcgaggtt gtttgttttg agattatagt tgttattttt tgtggttttt ttgatattta
60
ttttgatttt tttgttggtt aggttttttt tagtgatggt gttgttgtat ttattgtgg
119
<210> 73
<211> 119
<212> ДНК
<213> Искусственная последовательность
<220>
<223> CHSgu-R
<400> 73
gggtacctct agacaccatc aaaatctaaa aaaataatct aaacaacaaa caccatctca
60
aaaataaatt tctctccaac aaatatatca aaatccaacc ccacaataaa tacaacaac
119
<210> 74
<211> 120
<212> ДНК
<213> Искусственная последовательность
<220>
<223> asEIN2gu-F
<400> 74
caagcttaat gtttatgtga gttgtaatat attggtataa gatggtggta aaattgaaaa
60
gtagagtaat tgttattaag ttatatttga aatgagtatt ttttttgata tttgtaattt
120
<210> 75
<211> 120
<212> ДНК
<213> Искусственная последовательность
<220>
<223> asEIN2gu-R
<400> 75
gggatcctct agaacctcaa ctaaaattta tccaaaaaat aattcctact ctacttccta
60
tccttttaat ttctatcaaa tatattaatc ccaaaaaata aattacaaat atcaaaaaaa
120
<210> 76
<211> 120
<212> ДНК
<213> Искусственная последовательность
<220>
<223> asCHSgu-F
<400> 76
caagcttcta gagtattatt agagtttgga aggatggttt gagtggtaga tattatttta
60
aagatgggtt tttttttgat agatgtgtta gggtttgatt ttataatgag tgtggtggtg
120
<210> 77
<211> 120
<212> ДНК
<213> Искусственная последовательность
<220>
<223> asCHSgu-R
<400> 77
gggatccatt atctactcta aaatcacaac cattaccttc cataatccct ctaacaccca
60
ccttaactcc ctcatcaatc aaactctttt caataataac accaccacac tcattataaa
120
<210> 78
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> CHS-200-F2
<400> 78
gacatgcctg gtgctgacta
20
<210> 79
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> CHS-200-R2
<400> 79
ccttagcgat acggaggaca
20
<210> 80
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Actin2-For
<400> 80
tccctcagca cattccagca
20
<210> 81
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Actin2-Rev
<400> 81
gatcccattc ataaaacccc ag
22
<210> 82
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Top-35S-F2
<220>
<221> Y
<222> (8)..(8)
<223> C или T
<220>
<221> Y
<222> (11)..(11)
<223> C или T
<220>
<221> Y
<222> (14)..(14)
<223> C или T
<220>
<221> Y
<222> (17)..(17)
<223> C или T
<400> 82
agaaaatytt ygtyaayatg gtgg
24
<210> 83
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Top-35S-R2
<220>
<221> R
<222> (4)..(4)
<223> A или G
<220>
<221> R
<222> (6)..(7)
<223> A или G
<220>
<221> R
<222> (9)..(9)
<223> A или G
<220>
<221> R
<222> (12)..(12)
<223> A или G
<400> 83
tcartrrara trtcacatca atcc
24
<210> 84
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Link-35S-F2
<220>
<221> Y
<222> (1)..(2)
<223> C или T
<220>
<221> Y
<222> (5)..(5)
<223> C или T
<220>
<221> Y
<222> (10)..(10)
<223> C или T
<400> 84
yyatyattgy gataaaggaa agg
23
<210> 85
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Link-EIN2-R2
<220>
<221> R
<222> (6)..(6)
<223> A или G
<220>
<221> R
<222> (14)..(14)
<223> A или G
<400> 85
taattrccac caartcatac cc
22
<210> 86
<211> 22
<212> РНК
<213> Искусственная последовательность
<220>
<223> смысловой si22
<400> 86
gcaagcugac ccugaaguuc au
22
<210> 87
<211> 22
<212> РНК
<213> Искусственная последовательность
<220>
<223> антисмысловой si22
<400> 87
gaacuucagg gucagcuugc cg
22
<210> 88
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер
<400> 88
ttttagtata tgtgctgccg
20
<210> 89
<211> 83
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Обратный праймер
<400> 89
ctcgagttcc aaaaaagctg accctgaagt tcatctctct tgaagatgaa cttcagggtc
60
agccaaacaa ggcttttctc caa
83
<210> 90
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер
<400> 90
ttttagtata tgtgctgccg
20
<210> 91
<211> 83
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Обратный праймер
<400> 91
ctcgagttcc aaaaaaataa gtcgcagcag tacaatctct tgaattgtac tgctgcgact
60
tatgaatacc gcttcctcct gag
83
<210> 92
<211> 88
<212> РНК
<213> Искусственная последовательность
<220>
<223> молекулы дцРНК
<400> 92
aagugauuug ugaauggauu cgguuaaguu agugauaggu aucacgcugg ccauuacuga
60
caugaccgga uucguucgcg agucacuu
88
<210> 93
<211> 2653
<212> ДНК
<213> Brassica napus
<400> 93
agaaaaatcg aaaaatgtga cagtgcgtct tttcacttaa taccctcgtt ttgaatttgc
60
tctcggaaag cgtctgagag agtgttcggt gatttctccc gccgcttggg gttttttccg
120
ttaccggaat atccttctcc tccgatggtt agtctgcgct ccacagaaaa cactccggct
180
tcggaaatgg ccagcgacgg caaaacggag aaagatggct ccggcgactc acccacttct
240
gttctcagcg atgaggaaaa ctgtgaagag aaaactgcta ctgttgctgt agaggaagag
300
atacttctag ccaagaatgg agattcgtct cttatctctg aggccatggc tcaggaagaa
360
gagcagcttc tcaaaatccg ggaagatgaa gagattgcta aacgtgctgc tggctctggt
420
gaagctcctg atctgaatga tactcagttt actaaacttg atgagctctt gacccaaacc
480
cagctctact ctgagtttct ccttgagaaa atggaggata tcaccaaaaa tgggatagaa
540
ggtgagaccc aaaaggccga gcctgagcct gagcctgagc ccgagaagaa aggccgtgga
600
cgtaaaagaa aggctgctcc tcagggcgac agtatgaagg ctaagaaagc tgttgctgct
660
atgatttcaa gatccaaaga aggccgtgaa tctgccgact cagatctgac agaggaagaa
720
agagtcatga aagagcaggg tgaacttgtt cctcttctga ctggcggaaa gttaaagtct
780
tatcagctca aaggtgtcaa atggctgata tcattgtggc aaaatggttt gaatggaatt
840
ttagctgatc aaatgggtct tggaaagaca attcaaacca ttggtttcct atcacacctc
900
aaaggaaatg ggttggatgg tccatatcta gtcattgccc cactctctac tctttcaaac
960
tggatgaatg agatcgctag gttcacgcct tccattaatg caatcattta ccatggagat
1020
aagaaagaaa gggatgagct caggaagagg cacatgccca gaactgttgg tccgaagttc
1080
cctatagtca taacttctta tgaggttgct atgaatgatg ctaaaaagaa tctgcggcac
1140
tatccatgga aatatgttgt gattgatgag ggtcacaggt tgaaaaacca caagtgtaaa
1200
ctgctgaggg agctaagata cttgaatatg gagaacaaac ttctgctgcc aggaacacct
1260
ctgcaaaata atttgtctga gcttcggtca ctgttgaatt ttattctgcc tgacatcttt
1320
gcatcacatg acgaatttga atcatggttt gatttttctt gaaagaataa taatgaagca
1380
actaaggaag aaggagaaga gaaaagaaga gctcaagtgg ttgcgaaact tcataatata
1440
ctacgacctt tcatcctccg gagaatgaaa tgtgatgttg agctctcact tccccggaaa
1500
aaagagatta tcatctatgc tacaatgacg gaccatcaga agaagttcca ggaacatctt
1560
gtgaaccaca ccttggaagc acacattaga gatgatactg tccgaggtca tggcttgaag
1620
ggaaagctta acaatcttgc tattcaactt cgaaagaact gcaaccatcc tgaccttctt
1680
gtggggcaac tagatggctc atatctctac ccacctttgg aagacattgt gggacagtgc
1740
ggtaaattcc gcttattgga gagattgctt gttcggttat ttgccaaaaa tcacagagtc
1800
cttatcttct cccagtggac aaaaatactg gacattatgg attactactt cagtgagaag
1860
gggtttgagg tttgccgaat cgacggtagt gtgaaactag aagaaaggag aagacagatc
1920
caagaattca atgatgagaa gagcaactgc aggatatttc ttctcagtac cagagccgga
1980
ggactcggaa ttaatcttac tgctgcagat acatgcatcc tctacgatag cgattggaac
2040
cctcaaatgg acttgcaagc catggacaga tgccacagaa ttggtcagac aaaacctgtt
2100
catgtttaca ggcttgcgac ggctcagtca atagagggcc gagttctgaa acgagcatac
2160
agtaagctta agctggaaca tgtggttatt ggcaaggggc agtttcatca agaacgtgcc
2220
aagtcttcaa caccgttaga ggaagatgac atactggcgt tgcttaagga cgacgaaaat
2280
gctgaagata aactgataca aaccgacata agcgaggagg atcttgacag ggtgcttgac
2340
cgtagtgatc tgatgattac cttaccgggc gagactcaag cacatgaagc ttttccagtg
2400
aagggtccgg gttgggaagt ggtctcgtct agctcagctg gagggatgct gtcttccctc
2460
aacagttaga accactcttt gcaaaaccac ttcggtgtgt ttttttttcc ggaacataac
2520
cggttacttt tgcctgctac tcggaagttt taacttgaaa ccttggaaac atctgatgaa
2580
aacaattgcg gatattatgt tattagacta tttatttatg ccttttgaaa tttggcagta
2640
attttttagt taa
2653
<210> 94
<211> 1773
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая шпилечную РНКи (шРНК) конструкцию,
направленно воздействующую на ген DDM1 B. napus
<400> 94
gggagctgtt gctgctatga tttcaagatc caaagaaggc cgtgaatctg ccgactcaga
60
tctgacagag gaagaaagag tcatgaatga gcagggtgaa cttgttcctc ttctgactgg
120
cggaaagtta aagtcttatc agctcaaagg tgtcaaatgg ctgatatcat tgtggcaaaa
180
tggtttgaat ggaattttag ctgatcaaat gggtcttgga aagacaattc aaaccattgg
240
tttcctatca ccccaacaag gaaatgggtt ggatggtcca tatctagtca ttgccccact
300
ctctactctt tcaaaagcga ttggaaccct caaatggact tgcaagccat ggacagatgc
360
cacagaattg gtcagacaaa acctgttcat gtttacaggc ttgcgacggc tcagtcaata
420
gagggccgag ttctgaaacg agcatacagt aagcttaagc tggaacatgt ggttattggc
480
aaggggcagt ttcatcaaga acgtggtaag gaaataatta ttttcttttt tccttttagt
540
ataaaatagt taagtgatgt taattagtat gattataata atatagttgt tataattgtg
600
aaaaaataat ttataaatat attgtttaca taaacaacat agtaatgtaa aaaaatatga
660
caagtgatgt gtaagacgaa gaagataaaa gttgagagta agtatattat ttttaatgaa
720
tttgatcgaa catgtaagat gatatactag cattaatatt tgttttaatc ataatagtaa
780
ttctagctgg tttgatgaat taaatatcaa tgataaaata ctatagtaaa aataagaata
840
aataaattaa aataatattt ttttatgatt aatagtttat tatataatta aatatctata
900
ccattactaa atattttagt ttaaaagtta ataaatattt tgttagaaat tccaatctgc
960
ttgtaattta tcaataaaca aaatattaaa taacaagcta aagtaacaaa taatatcaaa
1020
ctaatagaaa cagtaatcta atgtaacaaa acataatcta atgctaatat aacaaagcgc
1080
aagatctatc attttatata gtattatttt caatcaacat tcttattaat ttctaaataa
1140
tacttgtagt tttattaact tctaaatgga ttgactatta attaaatgaa ttagtcgaac
1200
atgaataaac aaggtaacat gatagatcat gtcattgtgt tatcattgat cttacatttg
1260
gattgattac agtcacgttc ttgatgaaac tgccccttgc caataaccac atgttccagc
1320
ttaagcttac tgtatgctcg tttcagaact cggccctcta ttgactgagc cgtcgcaagc
1380
ctgtaaacat gaacaggttt tgtctgacca attctgtggc atctgtccat ggcttgcaag
1440
tccatttgag ggttccaatc gcttttgaaa gagtagagag tggggcaatg actagatatg
1500
gaccatccaa cccatttcct tgttggggtg ataggaaacc aatggtttga attgtctttc
1560
caagacccat ttgatcagct aaaattccat tcaaaccatt ttgccacaat gatatcagcc
1620
atttgacacc tttgagctga taagacttta actttccgcc agtcagaaga ggaacaagtt
1680
caccctgctc tttcatgact ctttcttcct ctgtcagatc tgagtcggca gattcacggc
1740
cttctttgga tcttgaaatc atagcagcaa cag
1773
<210> 95
<211> 1773
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая шпилечную РНКи (шРНК) конструкцию с
парами
оснований G:U, направленно воздействующую на ген DDM1 B. napus
<400> 95
gggttgttgt tgttatgatt ttaagattta aagaaggttg tgaatttgtt gatttagatt
60
tgatagagga agaaagagtt atgaatgagt agggtgaatt tgtttttttt ttgattggtg
120
gaaagttaaa gttttattag tttaaaggtg ttaaatggtt gatattattg tggtaaaatg
180
gtttgaatgg aattttagtt gattaaatgg gttttggaaa gataatttaa attattggtt
240
ttttattata ttttaaagga aatgggttgg atggtttata tttagttatt gttttatttt
300
ttattttttt aaaagtgatt ggaattttta aatggatttg taagttatgg atagatgtta
360
tagaattggt tagataaaat ttgtttatgt ttataggttt gtgatggttt agttaataga
420
gggttgagtt ttgaaatgag tatatagtaa gtttaagttg gaatatgtgg ttattggtaa
480
ggggtagttt tattaagaat gtgtggtaag gaaataatta ttttcttttt tccttttagt
540
ataaaatagt taagtgatgt taattagtat gattataata atatagttgt tataattgtg
600
aaaaaataat ttataaatat attgtttaca taaacaacat agtaatgtaa aaaaatatga
660
caagtgatgt gtaagacgaa gaagataaaa gttgagagta agtatattat ttttaatgaa
720
tttgatcgaa catgtaagat gatatactag cattaatatt tgttttaatc ataatagtaa
780
ttctagctgg tttgatgaat taaatatcaa tgataaaata ctatagtaaa aataagaata
840
aataaattaa aataatattt ttttatgatt aatagtttat tatataatta aatatctata
900
ccattactaa atattttagt ttaaaagtta ataaatattt tgttagaaat tccaatctgc
960
ttgtaattta tcaataaaca aaatattaaa taacaagcta aagtaacaaa taatatcaaa
1020
ctaatagaaa cagtaatcta atgtaacaaa acataatcta atgctaatat aacaaagcgc
1080
aagatctatc attttatata gtattatttt caatcaacat tcttattaat ttctaaataa
1140
tacttgtagt tttattaact tctaaatgga ttgactatta attaaatgaa ttagtcgaac
1200
atgaataaac aaggtaacat gatagatcat gtcattgtgt tatcattgat cttacatttg
1260
gattgattac agtcacgttc ttgatgaaac tgccccttgc caataaccac atgttccagc
1320
ttaagcttac tgtatgctcg tttcagaact cggccctcta ttgactgagc cgtcgcaagc
1380
ctgtaaacat gaacaggttt tgtctgacca attctgtggc atctgtccat ggcttgcaag
1440
tccatttgag ggttccaatc gcttttgaaa gagtagagag tggggcaatg actagatatg
1500
gaccatccaa cccatttcct tgttggggtg ataggaaacc aatggtttga attgtctttc
1560
caagacccat ttgatcagct aaaattccat tcaaaccatt ttgccacaat gatatcagcc
1620
atttgacacc tttgagctga taagacttta actttccgcc agtcagaaga ggaacaagtt
1680
caccctgctc tttcatgact ctttcttcct ctgtcagatc tgagtcggca gattcacggc
1740
cttctttgga tcttgaaatc atagcagcaa cag
1773
<210> 96
<211> 1260
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая конструкцию кпРНК, направленно
воздействующую на ген DDM1 B. napus
<400> 96
ggggtgatag gaaaccaatg gtttgaattg tctttccaag acccatttga tcagctaaaa
60
ttccattcaa accattttgc cacaatgata tcagccattt gacacctttg agctgataag
120
actttaactt tccgccagtc agaagaggaa caagttcacc ctgctctttc atgactcttt
180
cttcctctgt cagatctgag tcggcagatt cacggccttc tttggatctt gaaatcatag
240
cagcaacagc tttcttagcc ttcatactgt cgccctgagg agcagccttt cttttacgtc
300
cacggccttt cttctcgggc tcaggctcag gctcaggctc ggccttttgg gtctcacctt
360
ctatcccatt tttggtgata gctgttgctg ctatgatttc aagatccaaa gaaggccgtg
420
aatctgccga ctcagatctg acagaggaag aaagagtcat gaatgagcag ggtgaacttg
480
ttcctcttct gactggcgga aagttaaagt cttatcagct caaaggtgtc aaatggctga
540
tatcattgtg gcaaaatggt ttgaatggaa ttttagctga tcaaatgggt cttggaaaga
600
caattcaaac cattggtttc ctatcacccc aacaaggaaa tgggttggat ggtccatatc
660
tagtcattgc cccactctct actctttcaa aagcgattgg aaccctcaaa tggacttgca
720
agccatggac agatgccaca gaattggtca gacaaaacct gttcatgttt acaggcttgc
780
gacggctcag tcaatagagg gccgagttct gaaacgagca tacagtaagc ttaagctgga
840
acatgtggtt attggcaagg ggcagtttca tcaagaacgt cactacggtc aagcaccctg
900
tcaagatcct cctcgcttat gtcggtttgt atcagtttat cttcagcatt ttcgtcgtcc
960
ttaagcaacg ccagtatgtc atcttcctct aacggtgttg aagacttggc acgttcttga
1020
tgaaactgcc ccttgccaat aaccacatgt tccagcttaa gcttactgta tgctcgtttc
1080
agaactcggc cctctattga ctgagccgtc gcaagcctgt aaacatgaac aggttttgtc
1140
tgaccaattc tgtggcatct gtccatggct tgcaagtcca tttgagggtt ccaatcgctt
1200
ttgaaagagt agagagtggg gcaatgacta gatatggacc atccaaccca tttccttgtt
1260
<210> 97
<211> 4421
<212> ДНК
<213> Arabidopsis thaliana
<400> 97
tcttccaaaa tttgcccgcc attctctgtg tctcttcgtc taagggtttc ttccaaagaa
60
cgacgacaaa accactgaac ctaaaatccg aaatccaaaa gattcatgcg aaaaaatcgt
120
taaagagtac caatttcaga aagttacatt cttacagaga acaagtaatt ccccagaaat
180
gggatctagg gttccaatag aaaccatcga agaagacggc gaattcgatt gggaagcagc
240
agtcaaagaa atcgacttgg cttgtcttaa aaccacaaac gcttcttctt cttcgtcatc
300
ccatttcact cctttggcta atccaccaat tacggcaaat ctcactaagc cacctgcgaa
360
gagacaatct actctcgata aattcatcgg cagaaccgaa cataaaccgg agaatcatca
420
agttgtttcc gagtgtggtg ttaacgataa cgataatagt cctttagttg ggattgatcc
480
tgaggcagct aaaacttgga tttatccagt gaatgggagt gttcctttaa gagattatca
540
gtttgctata acgaagactg ctttgttttc gaatacattg gtggctttgc ctacgggact
600
tggtaaaacg cttatagctg cggttgttat gtataattac ttcagatggt ttccacaagg
660
taaaatagta tttgcggcgc cttctaggcc tcttgtgatg cagcagattg aggcgtgtca
720
taatattgtt ggaataccac aagaatggac gattgacttg acgggtcaga catgtccttc
780
gaaaagagct tttttgtgga aaagcaaacg ggttttcttt gtcactccac aagtgttaga
840
gaaggatata cagtcaggaa catgtcttac taactacttg gtttgcttgg tgatcgacga
900
ggcacatcga gctttaggga attattctta ttgtgttgta gttcgtgagt tgatggcggt
960
accgatacag ctgagaatac tggctcttac tgcaactcct ggatcaaaga cacaggccat
1020
ccagggtatc attgataatt tgcagatatc cacacttgaa tatcgaaatg agagtgacca
1080
tgatgtttgc ccttatgtcc acgacagaaa attagaagtc atcgaggttc ccttgggtca
1140
agatgcagat gatgtatcga aacgcctgtt tcatgttata cgtccatatg cagtcaggct
1200
taaaaacttt ggggttaatc taaatagaga tatacaaact ttaagtccac acgaagtact
1260
tatggcaagg gataagtttc gtcaagcacc tctaccaggc cttccccatg taaatcacgg
1320
agatgtagaa tcttgctttg cagctcttat cactctttat catattcgta agctcctttc
1380
tagtcatgga ataagaccag cgtatgagat gctagaagag aaattgaaag aagggccatt
1440
tgctaggttg atgagtaaga atgaagatat taggatgacg aagcttttga tgcagcaaag
1500
gttgtcacat ggagcaccaa gcccaaaatt gtcgaagatg ttagaaatac tggttgatca
1560
tttcaaagtg aaagatccga agacatcacg ggtcattatt ttctcaaatt tcagaggaag
1620
cgtaagagac ataatgaacg cattaagtaa tattggagat atggtcaaag caactgagtt
1680
tattggtcaa agttcaggta agacattgaa aggccagtcg caaaaaattc agcaggctgt
1740
tttggagaaa tttagagctg gggggttcaa tgttattgtc gcaacatcta ttggtgaaga
1800
aggcttggat atcatggaag ttgacctagt tatatgtttt gatgctaatg tatctcctct
1860
gaggatgatt caacggatgg gaagaactgg aaggaaaaat aatggtcgag ttgtagttct
1920
tgcttgtgaa ggatcagaaa agaacagcta tatgcgaaag caagcaagtg gacgggctat
1980
taaaaaacac atgcggaatg gaggaacaaa tagttttaat tttcatccta gtccaaggat
2040
gattccccat gtttataagc cagaagttca gcatgttgag ttttcaatca agcaattcgt
2100
tccacgtgga aagaaactac aagaggagta tgccactgag actccagctt tccagaaaaa
2160
gcttacacct gcagagacgc atatgctcgc taagtattac aacaaccccg atgaggaaaa
2220
gttgagagtg tccttaattg cgttccctca cttccagaca ttgccatcca aggtgcacaa
2280
agtaatgcat tcacgtcaaa caggcatgtt aattgacgct atgcagcact tgcaagagcc
2340
aactttttca gaacagagta aaagcttctt cactgagttt cgagctcctt tgggtgaaag
2400
agaagagctt gatacaggtc tgagggttac taatgatcca aaagatctac actctgtccg
2460
tgatttggaa gtcaacacat cacagagaaa ggcaaaacaa gttgaatctc ccacaagcac
2520
cttagagaca acagagaagg attacgaaga atcttcaccc acacaccgtt atcttttcag
2580
ttcagaatgt gcatccgttg atactctggg gaacgtcttc gtaatgccag ttcctctttt
2640
attctttcct aatgttctgg agtcagacaa tacgcctctg cctaaaacag aaaaacaaca
2700
ttcttgccgg aatacatctc acattgactt agttccagta gatacttcgg aaaaacatcg
2760
gcaagataat atctcatgca agttaaagga aagattctcg ccagacggtg ccagcgagac
2820
actagagact catagccttg tgaaaaggaa ctccaccaga gtaggtgaag atgatgtagc
2880
gaattctgtt ggagaaattg tgttatcatc ggatgaagat gactgtgagg gattggagct
2940
tagtccacgg ctcactaact tcatcaagag cggcattgtt ccagagtcac ctgtctatga
3000
ccaaggggaa gcgaacagag aagaagatct tgaatttcct cagctttctt cacccatgag
3060
gttcagtaac gaattggcag gagagtcttc tttccctgag agaaaggttc agcataagtg
3120
caacgattat aacattgtgt ctacaaccac tgaattgaga actcctcaga aggaggtagg
3180
tttggccaac ggaacagaat gcttggctgt ttctcctatt cctgaggatt ggagaactcc
3240
cttggcgaat ctgacaaaca caaacagcag cgctcgcaaa gattggcggg tgagttctgg
3300
agaaaagtta gaaactcttc gacagcctcg caagttgaag agactacgta gacttggaga
3360
ttgctcgagt gctgtaaagg agaattatcc tggtattaca gaggcagacc atatcagatc
3420
tcgttctcgc ggtaaaaagc acattagagg taagaagaag atgatcatgg atgatgatgt
3480
ccaagtcttc attgacgagg aagctgaggt ctcttcggga gcagagatgt cggctgatga
3540
gaacgaagat gtgactggcg attcatttga agatagtttc atagatgacg gaacaatgcc
3600
tacagcaaat actcaagccg agtctggtaa agttgacatg atggctgttt acaggcgttc
3660
tcttctcagc cagtcaccat taccggcgag atttcgtgat ttagccgcat caagtctgag
3720
tccttattct gctggaccct tgacgagaat aaatgagagc agaagcgact cagataaatc
3780
attgtcttct cttcgaacac caaaaacaac aaactctgag tcaaaccaag atgcaatgat
3840
gataggaaac ctttcggtag tacaaatctc gtcagatagc cggaaaagga aatttagctt
3900
atgcaactcg gcgaatgccc ccgtgattaa cttagaaagc aagtttgcag ctcatgcaca
3960
agccacggag aaggaaagcc atgaaggcgt gagaagcaat gcaggtgcgt tagagtacaa
4020
tgatgatgat gatgatgcat tctttgcgac actagacttt gatgcaatgg aagcacaagc
4080
cacattgtta ttgtcgaaac agagatccga agcaaaagag aaagaagacg caacggttat
4140
acctaatcca ggcatgcaga gaagtgatgg tatggagaaa gatgcaccat cttttgatct
4200
tggtctgtgg tgattcttct ttcatacgaa gatactaagt tatgtatata gattgacaaa
4260
ggagacagta gagcataggc atttggatgt atgttttgtg tattaagttt aggtatatcc
4320
tattgaagta cagtgcttaa ggcagtgcac atggttaaat caaggttaat gcctcaattc
4380
gttgaaccct ttaagtaatg acacaaatat gactacatcg g
4421
<210> 98
<211> 1771
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая шпилечную РНКи (шРНК) конструкцию,
направленно воздействующую на ген FANCM A. thaliana
<400> 98
gggtcaggaa catgtcttac taactacttg gtttgcttgg tgatcgacga ggcacatcga
60
gctttaggga attattctta ttgtgttgta gttcgtgagt tgatggcggt accgatacag
120
ctgagaatac tggctcttac tgcaactcct ggatcaaaga cacaggccat ccagggtatc
180
attgataatt tgcagatatc cacacttgaa tatcgaaatg agagtgacca tgatgtttgc
240
ccttatgtcc ccgacagaaa attagaagtc atcgaggttc ccttgggtca agatgcagat
300
gatgtatcga aacgcctgtt tcatgttata cgtccatatg cagtcaggct taaaaacttt
360
ggggttaatc taaatagaga tatacaaact ttaagtccac acgaagtact tatggcaagg
420
gataagtttc gtcaagcacc tctaccaggc cttccccatg taaatcacgg agatgtagaa
480
tcttgctttg cagctcttat caggtaagga aataattatt ttcttttttc cttttagtat
540
aaaatagtta agtgatgtta attagtatga ttataataat atagttgtta taattgtgaa
600
aaaataattt ataaatatat tgtttacata aacaacatag taatgtaaaa aaatatgaca
660
agtgatgtgt aagacgaaga agataaaagt tgagagtaag tatattattt ttaatgaatt
720
tgatcgaaca tgtaagatga tatactagca ttaatatttg ttttaatcat aatagtaatt
780
ctagctggtt tgatgaatta aatatcaatg ataaaatact atagtaaaaa taagaataaa
840
taaattaaaa taatattttt ttatgattaa tagtttatta tataattaaa tatctatacc
900
attactaaat attttagttt aaaagttaat aaatattttg ttagaaattc caatctgctt
960
gtaatttatc aataaacaaa atattaaata acaagctaaa gtaacaaata atatcaaact
1020
aatagaaaca gtaatctaat gtaacaaaac ataatctaat gctaatataa caaagcgcaa
1080
gatctatcat tttatatagt attattttca atcaacattc ttattaattt ctaaataata
1140
cttgtagttt tattaacttc taaatggatt gactattaat taaatgaatt agtcgaacat
1200
gaataaacaa ggtaacatga tagatcatgt cattgtgtta tcattgatct tacatttgga
1260
ttgattacag ttgataagag ctgcaaagca agattctaca tctccgtgat ttacatgggg
1320
aaggcctggt agaggtgctt gacgaaactt atcccttgcc ataagtactt cgtgtggact
1380
taaagtttgt atatctctat ttagattaac cccaaagttt ttaagcctga ctgcatatgg
1440
acgtataaca tgaaacaggc gtttcgatac atcatctgca tcttgaccca agggaacctc
1500
gatgacttct aattttctgt cggggacata agggcaaaca tcatggtcac tctcatttcg
1560
atattcaagt gtggatatct gcaaattatc aatgataccc tggatggcct gtgtctttga
1620
tccaggagtt gcagtaagag ccagtattct cagctgtatc ggtaccgcca tcaactcacg
1680
aactacaaca caataagaat aattccctaa agctcgatgt gcctcgtcga tcaccaagca
1740
aaccaagtag ttagtaagac atgttcctga c
1771
<210> 99
<211> 1771
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая шпилечную РНКи (шРНК) конструкцию с
парами
оснований G:U, направленно воздействующую на ген FANCM A.
thaliana
<400> 99
gggttaggaa tatgttttat taattatttg gtttgtttgg tgattgatga ggtatattga
60
gttttaggga attattttta ttgtgttgta gtttgtgagt tgatggtggt attgatatag
120
ttgagaatat tggtttttat tgtaattttt ggattaaaga tataggttat ttagggtatt
180
attgataatt tgtagatatt tatatttgaa tattgaaatg agagtgatta tgatgtttgt
240
ttttatgttt ttgatagaaa attagaagtt attgaggttt ttttgggtta agatgtagat
300
gatgtattga aatgtttgtt ttatgttata tgtttatatg tagttaggtt taaaaatttt
360
ggggttaatt taaatagaga tatataaatt ttaagtttat atgaagtatt tatggtaagg
420
gataagtttt gttaagtatt tttattaggt tttttttatg taaattatgg agatgtagaa
480
ttttgttttg tagtttttat taggtaagga aataattatt ttcttttttc cttttagtat
540
aaaatagtta agtgatgtta attagtatga ttataataat atagttgtta taattgtgaa
600
aaaataattt ataaatatat tgtttacata aacaacatag taatgtaaaa aaatatgaca
660
agtgatgtgt aagacgaaga agataaaagt tgagagtaag tatattattt ttaatgaatt
720
tgatcgaaca tgtaagatga tatactagca ttaatatttg ttttaatcat aatagtaatt
780
ctagctggtt tgatgaatta aatatcaatg ataaaatact atagtaaaaa taagaataaa
840
taaattaaaa taatattttt ttatgattaa tagtttatta tataattaaa tatctatacc
900
attactaaat attttagttt aaaagttaat aaatattttg ttagaaattc caatctgctt
960
gtaatttatc aataaacaaa atattaaata acaagctaaa gtaacaaata atatcaaact
1020
aatagaaaca gtaatctaat gtaacaaaac ataatctaat gctaatataa caaagcgcaa
1080
gatctatcat tttatatagt attattttca atcaacattc ttattaattt ctaaataata
1140
cttgtagttt tattaacttc taaatggatt gactattaat taaatgaatt agtcgaacat
1200
gaataaacaa ggtaacatga tagatcatgt cattgtgtta tcattgatct tacatttgga
1260
ttgattacag ttgataagag ctgcaaagca agattctaca tctccgtgat ttacatgggg
1320
aaggcctggt agaggtgctt gacgaaactt atcccttgcc ataagtactt cgtgtggact
1380
taaagtttgt atatctctat ttagattaac cccaaagttt ttaagcctga ctgcatatgg
1440
acgtataaca tgaaacaggc gtttcgatac atcatctgca tcttgaccca agggaacctc
1500
gatgacttct aattttctgt cggggacata agggcaaaca tcatggtcac tctcatttcg
1560
atattcaagt gtggatatct gcaaattatc aatgataccc tggatggcct gtgtctttga
1620
tccaggagtt gcagtaagag ccagtattct cagctgtatc ggtaccgcca tcaactcacg
1680
aactacaaca caataagaat aattccctaa agctcgatgt gcctcgtcga tcaccaagca
1740
aaccaagtag ttagtaagac atgttcctga c
1771
<210> 100
<211> 1259
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая конструкцию кпРНК, направленно
воздействующую на ген FANCM A. thaliana
<400> 100
gggacataag ggcaaacatc atggtcactc tcatttcgat attcaagtgt ggatatctgc
60
aaattatcaa tgataccctg gatggcctgt gtctttgatc caggagttgc agtaagagcc
120
agtattctca gctgtatcgg taccgccatc aactcacgaa ctacaacaca ataagaataa
180
ttccctaaag ctcgatgtgc ctcgtcgatc accaagcaaa ccaagtagtt agtaagacat
240
gttcctgact gtatatcctt ctctaacact tgtggagtga caaagaaaac ccgtttgctt
300
ttccacaaaa aagctctttt cgaaggacat gtctgacccg tcaagtcaat cgtccattct
360
tgtggtattc caacaatatg tcaggaacat gtcttactaa ctacttggtt tgcttggtga
420
tcgacgaggc acatcgagct ttagggaatt attcttattg tgttgtagtt cgtgagttga
480
tggcggtacc gatacagctg agaatactgg ctcttactgc aactcctgga tcaaagacac
540
aggccatcca gggtatcatt gataatttgc agatatccac acttgaatat cgaaatgaga
600
gtgaccatga tgtttgccct tatgtccccg acagaaaatt agaagtcatc gaggttccct
660
tgggtcaaga tgcagatgat gtatcgaaac gcctgtttca tgttatacgt ccatatgcag
720
tcaggcttaa aaactttggg gttaatctaa atagagatat acaaacttta agtccacacg
780
aagtacttat ggcaagggat aagtttcgtc aagcacctct accaggcctt ccccatgtaa
840
atcacggaga tgtagaatct tgctttgcag ctcttatcat tcgtcatcct aatatcttca
900
ttcttactca tcaacctagc aaatggccct tctttcaatt tctcttctag catctcatac
960
gctggtctta ttccatgact agaaaggagc ttacgaatat gataaagagt gataagagct
1020
gcaaagcaag attctacatc tccgtgattt acatggggaa ggcctggtag aggtgcttga
1080
cgaaacttat cccttgccat aagtacttcg tgtggactta aagtttgtat atctctattt
1140
agattaaccc caaagttttt aagcctgact gcatatggac gtataacatg aaacaggcgt
1200
ttcgatacat catctgcatc ttgacccaag ggaacctcga tgacttctaa ttttctgtc
1259
<210> 101
<211> 4228
<212> ДНК
<213> Brassica napus
<400> 101
tccaaaattg gttttgcccg ccaatgtggc ttcggcgagg gtttcttcca caaaacccca
60
ctcaacctaa aatctgattc ggcgagaaac gctgtctact tatctcacgc gaaaagaaag
120
gcgtagatcc accctaaact aaaacagagc atcaagtgaa atgggacccg agtttccgat
180
cgaactcgtt gaagaagaag atggattcga ttgggaagca gcagtcagag aaatcgactt
240
ggcttgcctc aaatccttaa acccttcttc ttcttcttcg acccatttca ccaacggcaa
300
tggcactaaa cctgctaaaa gacaatctac tcttgatcga ttcatcgcaa gagccgacca
360
caagcctcct cctccgtatc ctcctgttgt ttccgacccg agtttcgagt gtggtactaa
420
cgacaacact cccagcgtcg ggattgatcc tgagacagct aaaacttgga tttatccaat
480
gaacgttcct ctaagagatt atcagtttgc tataacgaag actgctttgt tttcaaacac
540
attagttgct ttaccaacag gccttggtaa aacgctcata gctgcagttg taatgtataa
600
ttacttcaga tggtttccac aaggtaaaat tgtctttgcc gcaccttcta ggcctcttgt
660
gatgcagcag attgaggcct gccataatat cgtggggata ccacaagaat ggacgattga
720
cttgacgggt cagacttgcc cttccaaaag agcttccttg tggaaaacca aaagggtttt
780
cttcgtcact ccacaagttc ttgagaagga tatacagtca ggaacgtgtg ttaccaactg
840
cttggtttgc ttggtgatcg acgaggcaca tcgagcttta gggaattatt cttattgtgt
900
tgtagttcgt gagttgatgg cagtaccagt gcagttgaga atattggctc ttactgcaac
960
tcctggatca aagacacagg ccatacaggg tatccttgat aatttgcaga tatcaacact
1020
tgaatatcga aacgagagtg accatgatgt ctgcccttat gtccacgaca gaaaagtaga
1080
actaatcgag gttcccttgg gtaaagatgc agatgaggta tctaaacgcc tattagatgt
1140
tatacgtcca tatgctgtca ggcttaaaaa tttcggggtc attctaagca gggattatca
1200
aactttgagt ccacacgaat tacttatggc aagggataag tttcgtgaag cacctgtacc
1260
aggcattccc catataagtc acggagatgt agaatcttgc tttgcagctc ttatcacgct
1320
ttatcacatt cgcaagcttc tttctagtca tggaataagg ccagcgtatg agatgcttga
1380
agaaaaactt caggaagggc catttgctag gttgatgagt aagaatgaag atattaggat
1440
gacgaagctt ttgatgcagc aaaggttgtc gaacggagca ccaagcccga aattgtccaa
1500
gatgttggag attctagttg atcactacaa aataaaagat ccgaggacat cacgggtcat
1560
tattttctcg aatttcagag gaagcgtaag agacataatg gacgcattaa gtaatattga
1620
agatgttgtc aaagcaactg agtttattgg tcaaagttca ggtaagacac tgaagggaca
1680
gtcgcaaaaa gttcagcaag ctgttctgga gaaatttaga tctggtgggt ttaatgttat
1740
tgttgcaaca tctatcggcg aagaaggctt ggatatcatg gaagtcgact tagttatatg
1800
ttttgatgct aatgtatccc ctctgaggat gatccaacgc atgggaagaa ctggaaggaa
1860
aaataatggc cgagttgtag ttcttgcttg tgaaggatct gaaaagaata gctatatgcg
1920
aaagaaagca aatggccaag ccattaaaaa acacatgcgg aatggaggaa tgaatagttt
1980
taattttcat cctagtccaa ggatgattcc ccatgtttat aagccagaag ttcagcatgt
2040
taagttttcg atcgagcaat tcattccacg tggaaagaag ctacaagatg agcctgccac
2100
tgagactcca gctttcaaga aaaagcttac accggaagag atggatatgc tcgccaagta
2160
tttcaaaccc aacgaggaaa agtggagagt ttccttgatt gctttccctc acttccaaac
2220
attgccatcc aaagtgcaca aagtaatgca ttcacgccaa acaagcatat taattgatgc
2280
tatgcagcat ctgcaagaga caactttgac agagcaaagt aaaagtttct tcattaagta
2340
tggagctcct ttggctgaaa gagatgagct tgacgcaggt ctgagggttg gtgatgatcc
2400
gaaaggtaaa tttagtctca atgatttgga tggcaacaca tcacagagaa aggcaaaaca
2460
aattttagaa tctcccacaa gcacattaga gactacagag aaggatttcg aagcatcttc
2520
acccacacac tgttatcttt tcagttcaga atgtgcgtcc gttgatactc tggggaaggt
2580
ctttgtattg ccggttcctc tctcattctc ttctaatgta ccagggtcag actgcgtggg
2640
aagagaaaaa gaactttctt ccccgaataa gtcccacact gacgttgttc cgatagatag
2700
ttcctcaaaa catcggcaag ataatatttc atgcaagtta aagcaaggat tcttgccaga
2760
ttgtgccaac gagactttgg agtcccaaag ccttttgaaa aggcactcca ccgatgtagg
2820
taaaggagat atagagaatt gtgctggaga aattatgata tcatcggatg aagaagacga
2880
ctgtgaggat ttggagctta gtccaaggct cactaacttc atcaagagtg gcgttgttcc
2940
agattcacct gtctatgacc aaggagttgc atacgaagca aacagagaag aagaccttga
3000
tcttccaccc acgagtttaa ctaatgaatt ggcagaagag ccatcgacac ctgagaaaaa
3060
ggttcacatt gcttctacgg ccaatgaatt cagaactcct cagaaggaag aagatttagc
3120
caacgaaaca gaaagcttcg ctgtttctcc aatgcctgag gagtggagaa ctcccttggc
3180
gaatatcacc aacgcaagca gcagcgctag caaagattgg cgcgtgagtt cgggagaaaa
3240
gtcagaaact cttcgacagc ctcgcaagtt gaagagactt cgtagacttg gagattgctc
3300
gagtgctgtg aaggagaata atcctggtat tgcaaagaca gaccatatca gatctcgttc
3360
tcgcagtgta aagaacataa gaggcaagaa gaagatacgc gcggataata atgctagaat
3420
cttcattgaa gcggaagctg aggtgtcttc ggaatcagaa atgtcggttg atgagaacgt
3480
agatttgacc agcgattcat ttgaagatag cttcatagat gacggtacaa tgcctacagc
3540
aaatactcaa gccgagtgtg ctaaagttga catgatggcc gtttacagac gttctctact
3600
cagccaatca ccattaccgg caagatttcg tgatgtagct gcatcaagtc cgagtcctta
3660
ttcttctggt ctcttgaaga caataaatga gagcagaagc gactcagata aatcattgtc
3720
ttctcttaga accccacaaa caacgaacaa cgagtcaaac aaggatgcag tggccacagg
3780
agactttttg gtagcacaaa tctcaacaga cagccggaaa aggaaattca gcttatgcaa
3840
ctcagcgaat gtcccagtga ttaacttgga aaacaagttt gaagctcatg cacaagccac
3900
ggagaaggaa agccatgaag gtccgagaag caatgcaggt gcatcacagt acaaggatga
3960
ggatgaagat gatgatgcat tctacgcgac actggacttt gatgccatgg aagcgcatgc
4020
gacattgcta ttgtcgaaac aaaggtcaga aacgaaaaca aaagaagatg catcggtgaa
4080
acctcatttg ggcaatcaga ggaatgatgg tttgccgaag gatgggccat cttttgatct
4140
tggtttgtgg tgattattct cctattaagt taaagtgtat aaaggttgac atttggatgt
4200
atgttttgtg tatttagttt gtgtcata
4228
<210> 102
<211> 1769
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая шпилечную РНКи (шРНК) конструкцию,
направленно воздействующую на ген FANCM B. napus
<400> 102
gggagaaatt atgatatcat cggatgaaga agacgactgt gtggatttgg agcttagtcc
60
aaggctcact aacttcatca agagtggcgt tgttccagat tcacctgtct atgaccaagt
120
tgcatacgaa gcaaacagtg aagaagacct tgatcttcca cacacgagtt taactaatga
180
attggcagaa gagccatcga cacctgagaa aaaggttcac attgcttcta cggccaatga
240
attcagaacc ccaacgaagg aagaagattt agccaacgaa acagaaagct tcgctgtttc
300
tccaatgcct gaggagtgga gaactccctt ggcgaatatc accaacgcaa gcagcagcgc
360
tagcaaagat tggcgcgtga gttcgggaga aaagtcagaa actcttcgac agcctcgcaa
420
gttgaagaga cttcgtagac ttggagattg ctcgagtgct gtgaaggaga ataatcctgg
480
tattgcaaag acagaccata tcgtaaggaa ataattattt tcttttttcc ttttagtata
540
aaatagttaa gtgatgttaa ttagtatgat tataataata tagttgttat aattgtgaaa
600
aaataattta taaatatatt gtttacataa acaacatagt aatgtaaaaa aatatgacaa
660
gtgatgtgta agacgaagaa gataaaagtt gagagtaagt atattatttt taatgaattt
720
gatcgaacat gtaagatgat atactagcat taatatttgt tttaatcata atagtaattc
780
tagctggttt gatgaattaa atatcaatga taaaatacta tagtaaaaat aagaataaat
840
aaattaaaat aatatttttt tatgattaat agtttattat ataattaaat atctatacca
900
ttactaaata ttttagttta aaagttaata aatattttgt tagaaattcc aatctgcttg
960
taatttatca ataaacaaaa tattaaataa caagctaaag taacaaataa tatcaaacta
1020
atagaaacag taatctaatg taacaaaaca taatctaatg ctaatataac aaagcgcaag
1080
atctatcatt ttatatagta ttattttcaa tcaacattct tattaatttc taaataatac
1140
ttgtagtttt attaacttct aaatggattg actattaatt aaatgaatta gtcgaacatg
1200
aataaacaag gtaacatgat agatcatgtc attgtgttat cattgatctt acatttggat
1260
tgattacagg atatggtctg tctttgcaat accaggatta ttctccttca cagcactcga
1320
gcaatctcca agtctacgaa gtctcttcaa cttgcgaggc tgtcgaagag tttctgactt
1380
ttctcccgaa ctcacgcgcc aatctttgct agcgctgctg cttgcgttgg tgatattcgc
1440
caagggagtt ctccactcct caggcattgg agaaacagcg aagctttctg tttcgttggc
1500
taaatcttct tccttcgttg gggttctgaa ttcattggcc gtagaagcaa tgtgaacctt
1560
tttctcaggt gtcgatggct cttctgccaa ttcattagtt aaactcgtgt gtggaagatc
1620
aaggtcttct tctctgtttg cttcgtatgc aacttggtca tagacaggtg aatctggaac
1680
aacgccactc ttgatgaagt tagtgagcct tggactaagc tccaaatcct cacagtcgtc
1740
ttcttcatcc gatgatatca taatttctc
1769
<210> 103
<211> 1769
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая шпилечную РНКи (шРНК) конструкцию с
парами
оснований G:U, направленно воздействующую на ген FANCM B.
napus
<400> 103
gggagaaatt atgatattat tggatgaaga agatgattgt gtggatttgg agtttagttt
60
aaggtttatt aattttatta agagtggtgt tgttttagat ttatttgttt atgattaagt
120
tgtatatgaa gtaaatagtg aagaagattt tgatttttta tatatgagtt taattaatga
180
attggtagaa gagttattga tatttgagaa aaaggtttat attgttttta tggttaatga
240
atttagaatt ttaatgaagg aagaagattt agttaatgaa atagaaagtt ttgttgtttt
300
tttaatgttt gaggagtgga gaattttttt ggtgaatatt attaatgtaa gtagtagtgt
360
tagtaaagat tggtgtgtga gtttgggaga aaagttagaa attttttgat agttttgtaa
420
gttgaagaga ttttgtagat ttggagattg tttgagtgtt gtgaaggaga ataattttgg
480
tattgtaaag atagattata ttgtaaggaa ataattattt tcttttttcc ttttagtata
540
aaatagttaa gtgatgttaa ttagtatgat tataataata tagttgttat aattgtgaaa
600
aaataattta taaatatatt gtttacataa acaacatagt aatgtaaaaa aatatgacaa
660
gtgatgtgta agacgaagaa gataaaagtt gagagtaagt atattatttt taatgaattt
720
gatcgaacat gtaagatgat atactagcat taatatttgt tttaatcata atagtaattc
780
tagctggttt gatgaattaa atatcaatga taaaatacta tagtaaaaat aagaataaat
840
aaattaaaat aatatttttt tatgattaat agtttattat ataattaaat atctatacca
900
ttactaaata ttttagttta aaagttaata aatattttgt tagaaattcc aatctgcttg
960
taatttatca ataaacaaaa tattaaataa caagctaaag taacaaataa tatcaaacta
1020
atagaaacag taatctaatg taacaaaaca taatctaatg ctaatataac aaagcgcaag
1080
atctatcatt ttatatagta ttattttcaa tcaacattct tattaatttc taaataatac
1140
ttgtagtttt attaacttct aaatggattg actattaatt aaatgaatta gtcgaacatg
1200
aataaacaag gtaacatgat agatcatgtc attgtgttat cattgatctt acatttggat
1260
tgattacagg atatggtctg tctttgcaat accaggatta ttctccttca cagcactcga
1320
gcaatctcca agtctacgaa gtctcttcaa cttgcgaggc tgtcgaagag tttctgactt
1380
ttctcccgaa ctcacgcgcc aatctttgct agcgctgctg cttgcgttgg tgatattcgc
1440
caagggagtt ctccactcct caggcattgg agaaacagcg aagctttctg tttcgttggc
1500
taaatcttct tccttcgttg gggttctgaa ttcattggcc gtagaagcaa tgtgaacctt
1560
tttctcaggt gtcgatggct cttctgccaa ttcattagtt aaactcgtgt gtggaagatc
1620
aaggtcttct tctctgtttg cttcgtatgc aacttggtca tagacaggtg aatctggaac
1680
aacgccactc ttgatgaagt tagtgagcct tggactaagc tccaaatcct cacagtcgtc
1740
ttcttcatcc gatgatatca taatttctc
1769
<210> 104
<211> 1259
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая конструкцию кпРНК, направленно
воздействующую на ген FANCM B. napus
<400> 104
gggttctgaa ttcattggcc gtagaagcaa tgtgaacctt tttctcaggt gtcgatggct
60
cttctgccaa ttcattagtt aaactcgtgt gtggaagatc aaggtcttct tctctgtttg
120
cttcgtatgc aacttggtca tagacaggtg aatctggaac aacgccactc ttgatgaagt
180
tagtgagcct tggactaagc tccaaatcct cacagtcgtc ttcttcatcc gatgatatca
240
taatttctcc agcacaattc tctatatctc ctttacctac atcggtggag tgccttttca
300
aaaggctttg ggactccaaa gtctcgttgg cacaatctgg caagaatcct tgctttaact
360
tgcatgaaat attatcttgg agaaattatg atatcatcgg atgaagaaga cgactgtgtg
420
gatttggagc ttagtccaag gctcactaac ttcatcaaga gtggcgttgt tccagattca
480
cctgtctatg accaagttgc atacgaagca aacagtgaag aagaccttga tcttccacac
540
acgagtttaa ctaatgaatt ggcagaagag ccatcgacac ctgagaaaaa ggttcacatt
600
gcttctacgg ccaatgaatt cagaacccca acgaaggaag aagatttagc caacgaaaca
660
gaaagcttcg ctgtttctcc aatgcctgag gagtggagaa ctcccttggc gaatatcacc
720
aacgcaagca gcagcgctag caaagattgg cgcgtgagtt cgggagaaaa gtcagaaact
780
cttcgacagc ctcgcaagtt gaagagactt cgtagacttg gagattgctc gagtgctgtg
840
aaggagaata atcctggtat tgcaaagaca gaccatatcc agcttccgct tcaatgaaga
900
ttctagcatt attatccgcg cgtatcttct tcttgccttg aacacagagc aaaaggaaat
960
acagaatcat tttacctctt atgttcttta cactgcgaga acgagatctg atatggtctg
1020
tctttgcaat accaggatta ttctccttca cagcactcga gcaatctcca agtctacgaa
1080
gtctcttcaa cttgcgaggc tgtcgaagag tttctgactt ttctcccgaa ctcacgcgcc
1140
aatctttgct agcgctgctg cttgcgttgg tgatattcgc caagggagtt ctccactcct
1200
caggcattgg agaaacagcg aagctttctg tttcgttggc taaatcttct tccttcgtt
1259
<210> 105
<211> 7074
<212> ДНК
<213> Nicotiana benthamiana
<400> 105
atgcgagctg tttttgaaac aaagcgtgat cgtgaaatct tggtccttgc tagtaaagtt
60
ctgggtcacc tagctagatc tggcggtgca atgactgcag atgaagtgga acgtcagata
120
aaagttgcac taggatggct tcgtggtgaa agaattgagt atcgtttctt tgctgctgtc
180
ttaatattga aggaaatggc ggaaaatgct tcaactgttt tcaatgttca tgtgccggac
240
tttgtggagg ttgtttgggt tgctctgaag gatccaacat tggctgttcg agagaaggct
300
gttgaggcat tgcgtgcctg ccttcgcgtt attgaaaagc gcgagacacg atggcgtgtt
360
cagtggtatt ataagatgtt tgaggctacc caagatggat tgaccagaac tgcgcctgtt
420
catagtatac atggctccct tctcgcagtg ggagagctgc taaggaatac aggagagttc
480
atgatgtcaa gatacaggga ggttgcagaa attgttataa gatacttgga gcaccgagat
540
cgcctagttc ggctcagcat aacttctcta cttcctcgaa ttgctcattt cctgcgtgat
600
cgatttgtga ctaactactt aacgatatgc atgaatcata tacttcatgt ccttaaaata
660
cctgcagaac gtgccagtgg gttcattgct cttggggaga tggctggtgc tctggatggt
720
gaactcatta actatttgcc gacaataacc tctcacttgc gtgatgcgat tgctccccgt
780
agaggcaggc cctcatttga ggctctggca tgtgttggaa atattgctaa agcaatggga
840
cctgcaatgg agcctcatgt tcgtggtctc ttggatgcta tgttttctgc tgggctttcc
900
ctgacactag tggaagcctt ggagcaaata actgaaagca ttccatcttt gttgccgacc
960
attcaagatc ggcttcttga atgtatttca gcaattctct ccagatctaa tcatgcactc
1020
tcaagacaat caactgctat gagtcgagga catattgcaa cagttacccc ccaagtacca
1080
gaactgagtg gtcctgcact agttcaactt gctttgcaga ctcttgctcg ttttaatttc
1140
aagggccatg atcttcttga gtttgcaagg gagtctgttg ttgtgtattt agaagatgag
1200
gatggagcta cacgaaaaga tgctgcgcta tgttgctgca aactagtagc aaattctttc
1260
ttggcgatat cttctaccca gtttagtcct agtagaatca atcgtgccag tggaaagcga
1320
cgtcgacttg ttgaagagat tgtgcaaaaa cttctcatcg ctgctgttgc cgacgctgat
1380
gttactgttc ggcattcaat tttttcttct ctgtatgctg atggaggatt cgatgagttt
1440
ctagctcagg ctgatagttt gacagctata tttgccactt taaatgatga ggattttgaa
1500
gttcgtgact atgcaatttc actagctggt agactatctg aaaagaatcc agcatatgtt
1560
cttccagcac ttcgtcgcca tcttattcag ctgttaactt acctagagca aagtgcagat
1620
aataaatgta aagaagagag tgcaaagtta ttgggttgct tgattcgcaa ttgtgaacga
1680
ttagttcttc catacattgc tcccatacac aaggctcttg ttgcgaaact ctgtgaaggc
1740
acaggagtca atgcgaatag tggcattatt agtggagttc tagtgactgt tggagatctt
1800
gccagagtgg gtggctttgc catgcggcag tatatttcag aacttatgcc attaatcgtt
1860
gaagctctac tggatggggc agctgccacc aaacgtgaag tggccgtttc aacacttggt
1920
caagttgtac agagtacagg atatgtcata actccataca atgagtatcc tcagttgctt
1980
ggtttactct tgaaactgct caatggtgaa ctggcttggt caaccagaag agaggttttg
2040
aaggttctcg gcatcatggg tgcattagat ccccatgtgc acaagcgcaa tcagcaaagc
2100
ttacccggat cccatggtga agttacccgg gtgactggtg atcctggtca acatatcaga
2160
tcaatggatg aattgcctat ggatctttgg ccctcctttg caacatctga agattattat
2220
tccactgttg ctatcaactc actcatgcgg atactcaggg atccatctct gtcaagttac
2280
caccagaaag tggttggatc tcttatgttt attttcaagt ccatgggcct tggctgtgtc
2340
ccttatttgc ctaaggtttt gcctgatctc tttcacattg tacgaacatg tgaggatggt
2400
ctaaaagaat ttataacatg gaagcttgga accttggtat ctattgtccg ccagcacatc
2460
cgtaagtatc tgccagagtt actctctctg atatcagaaa tatggtcatc tttcagcttg
2520
cctgttgcta acagacctgt tcacattgct cctattttgc atctcgtgga gcaactttgc
2580
ttggctctca acgatgaatt tagaaagtac cttgctgata tacttccctg ctgtattcaa
2640
gttcttactg atgcagagag gtttagtgac tacacatacg ttattcctat tctccacaca
2700
cttgaagttt ttggtgggac attagatgag catatgcatc tgcttcttcc tgcacttatt
2760
cggttgttta aattggatgc ttcagtagaa gtaagacgcg gtgcaatcaa aactctcaca
2820
agattgatac ctcgtgtgca ggtcactgga cacatatctt ctcttgtgca tcacttgaag
2880
cttgtcttgg acgggaacaa agaagagctc aggaaggatg ctgttgatgc actttgttgt
2940
ctagctcatg ctcttggaga ggacttcacc atttttattc attctattca caagcttatg
3000
gttaaacata ggctgcagca caaggaattt gaagaaatcc gaggacgact ggaaaaacgt
3060
gagccactga ttttggggag caccgcagct cagagattaa atcggcggtt cccagttgag
3120
gtcatcagtg atcctttgag tgatggagag aatgagcact acgaggttgg gacggacatg
3180
cataagcagc ttaaaagcca tcaggttaat gatggtagat tgcgtaccgc tggtgaggct
3240
tctcaacgaa gcactaaaga ggattgggca gagtggatga ggcatttcag cattgaactt
3300
ctgaaagaat cacctagtcc agcattgcga acttgtgcaa gactcgctca actgcagcct
3360
tttgttgggc gagagttgtt tgctgcaggt tttgttagct gctggtcaca acttaatgag
3420
gctagtcaaa ggcagctagt acgtagtcta gaaatggcat tttcgtctcc aaatatccct
3480
cctgaaattc ttgctacact tctgaacttg gcggagttta tggaacacga tgagagaccc
3540
cttcctattg atatccgtct gcttggtgct cttgcggaga agtgtcgagc atttgcaaag
3600
gccctacact acaaggaaat ggaatttgaa ggcgcacttt caaataggag ggatgcaaat
3660
cctgttgctg tagttgaagc tctaatccat ataaataatc aattacatca acatgaggca
3720
gctgttggaa tattaacata tgctcagcag catttggggg ttcaattgaa ggagtcatgg
3780
tacgagaaat tgcaacgctg ggatgatgct cttaaagcat acactgctaa ggcgtcacaa
3840
gcttcgagtc cacatcttgc tttggatgct actttagggc gtatgcgatg ccttgctgct
3900
ctagctcggt gggaggagct taacaatctt tgtaaggaat actggacacc agctgagcca
3960
gcagctcgac tggaaatggc accaatggct gctagtgcgg cctggaacat gggtgagtgg
4020
gatcagatgg cagagtatgt ttctcggctt gatgatggtg atgaaaccaa actgcgagtc
4080
ttgggaaata ccgctgccag tggcgatgga agtagtaatg gcaccttttt cagggctgtt
4140
cttctagttc ggcgagggaa gtacgatgaa gcacgtgaat atgttgaaag agcaaggaaa
4200
tgtttggcga ccgagctcgc tgcactggtt cttgagagct atgaacgtgc ttacagcaac
4260
atggtccgtg ttcagcagct ttctgaatta gaagaggtga ttgaatactg tactcttcct
4320
atgggaaacc ctgttgctga aggaagaaga gctcttgttc gcaatatgtg gaatgagcgc
4380
ataaagggta caaaaagaaa tgttgaggtt tggcaagtac ttttagctgt gagggcactt
4440
gtattgcctc ctacagaaga cattgaaaca tggatcaaat ttgcatcact ttgccggaag
4500
aatggcagaa ttagccaagc tagatctaca ttggttaaac ttttacagtt cgatccagaa
4560
tcaactcctg caactgtgcg gtatcatggt ccccctcagg tgatgctagc atacttaaag
4620
taccaatggt cacttggcga ggatcataag cgaaaggaag cctttgctag gttgcaggac
4680
cttgccatgg acctctcaag aacagcagct cttcaaccag tattgcagaa tggattagtt
4740
gcttcttctg gtgtgccact tgttgctcgt gtatatctca gactcggcac ttggaagtgg
4800
gcactttctc ctggtttgga tgatgattct attcaagaaa ttcttagtgc atttacaaat
4860
gctactcact gtgcaacgaa gtggggaaag gcatggcata cctgggcact tttcaatacc
4920
gcagtgatgt ctcattacac tctgagaggt tttgcgaata ttgcttcaca gtttgttgtt
4980
gctgccgtaa ctggttattt tcactctata gcatgcggag cacatgctaa gggtgttgat
5040
gatagtttac aggatattct tcgtcttctt actttgtggt tcaaccatgg agctacttcg
5100
gatgtccaaa tggcattgca gaaaggattc actcatgtta acatcaacac atggttggtt
5160
gttttacctc agattattgc acggatacat tcaaataacc atgctgtcag agaactgata
5220
caatccttgc tagtgcgaat tggacagagt catccacagg ctcttatgta tccgcttctt
5280
gtggcatgta agtcaattag caatttgcgc agagctgcgg ctcaagaagt ggttgataaa
5340
gttagacagc acagcggcgt actcgttgat caggcccaac ttgtctcaaa ggagcttatc
5400
agggttgcaa tactgtggca tgaaatgtgg catgaggcac tggaagaggc cagccgttta
5460
tattttggcg aacacaacat tgagggcatg ctgaaggtgt tagagcctct gcatgaaatg
5520
cttgaggaag gagcgatgag gaacaatacc actataaagg agaaagcatt catccaggca
5580
taccgtcttg agttgttgga ggcgtatgaa tgttgtatga agtatcggag aactggtaaa
5640
gatgctgaat taacgcaggc ttgggatctc tattatcatg tattcaggcg gatagataag
5700
cagcttcaaa cactcacaac cctggatttg cagtctgttt cccccgagtt actggagtgt
5760
cgaaatttgg agctagctgt tcctggaact tatatagcag atgcaccagt ggtgacaatt
5820
gcatcatttg caccccaact tgttgtaatt acatccaaac aacggcctcg aaaattgaca
5880
atccatggga gtgatggaga agactatgct tttttgctca aagggcacga agatctacgc
5940
caagatgaac gtgtcatgca gttgtttggt ctggttaata ctttgctcga gaattcaaga
6000
aagactgcag agaaagattt atcaattcaa cgatatgctg tcattccatt gtcccctaat
6060
agtggactga taggatgggt tccaaattgc gacaccttgc accagcttat tcgagaatat
6120
agggatgccc ggaagatcac cctaaatcaa gagcataaat tgatgctgag ttttgcaccg
6180
gattatgata atttgccact tattgctaag gtggaggtgt ttgaatatgc tttgcaaaat
6240
acagaaggga atgacttatc aagggttctt tggttaaaga gtcgtacttc tgaagtctgg
6300
ctggacagaa gaacaaatta tacaagaagt ttggctgtca tgagtatggt tggataccta
6360
cttggtctgg gtgatcgaca tcctagtaac ctcatgcttc accgatacag tgggaagatt
6420
ctgcatattg actttggaga ttgctttgaa gcttcaatga atcgggagaa gtttccagag
6480
aaggttccct ttcgactcac tagaatgctt gtaaaagcaa tggaggttag tggtatagag
6540
ggaaatttcc ggtcaacatg tgagaatgta atgcaagttc tccgactgca taaagatagt
6600
gttatggcta tgatggaggc ctttgttcac gatccactta taaattggcg tcttttcaac
6660
ttcaatgaag ttccgcaaat gtccgcactt gccagtgcac atgtccctcc tgttgtgaac
6720
agtgaggaat cttcttcaaa tagagagctt cttcagccac aaaggggtgc aagggagaga
6780
gaactgcttc aggcggtcaa tcaattaggt gatgccaatg aggttctaaa tgaacgtgct
6840
gtggctgtta tggctcgaat gagtaataaa ctcacaggac gtgattttgc tgctacttct
6900
acatctgcga gctctctaca acatgcactg gaccacagta cgttaatttc tggagagacg
6960
cgtgaagctg atcatggttt atcagtgaaa ctacaagtcc aaaaacttat tcaacaagcg
7020
tcgtctcatg aaaatctttg ccaaaattat gttgggtggt gtccattttg gtag
7074
<210> 106
<211> 1513
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая конструкцию кпРНК, направленно
воздействующую на ген TOR N. benthamiana
<400> 106
tgaatttagg tgacactata gaacaaagaa gagctcagga aggatgctgt tgatgcactt
60
tgttgtctag ctcatgctct tggagaggac ttcaccattt ttattcattc tattcacaag
120
cttatggtta aacataggct gcagcacaag gaatttgaag aaatccgagg acgactggaa
180
aaacgtgagc cactgatttt ggggagcacc gcagctcaga gattaaatcg gcggttccca
240
gttgaggtca tcagtgatcc tttgagtgat ggagagaatg agcactacga ggttgggacg
300
gacatgcata agcagcttaa aagccatcag gttaatgatg gtagattgcg taccgctggt
360
gaggcttctc aacgaagcac taaagaggat tgggcagagt ggatgaggca tttcagcatt
420
gaacttctga aagaatcacc tagtccagca ttgcgaactt ttaagctgct tatgcatgtc
480
cgtcccaacc tcgtagtgct cattctctcc atcactcaaa ggatcactga tgacctcaac
540
tgggaaccgc cgatttaatc tctgagctgc ggtgctcccc aaaatcagtg gctcacgttt
600
ttccagtcgt cctcggattt cttcaaattc cttgtgctgc agcctatgtt taaccataag
660
cttgtgaata gaatgaataa aaatggtgaa gtcctctcca agagcatgag ctagacaaca
720
aagtgcatca acagcatcct tcctgagctc ttctttgttc ccgtccaaga caagcttcaa
780
gtgatgcaca agagaagata tgtgtccagt gacctgcaca cgaggtatca atcttgtgag
840
agttttgatt gcaccgcgtc ttacttctac tgaagcatcc aatttaaaca accgaataag
900
tgcaggaaga agcagatgca tatgctcatc taatgtccca ccaaaaactt caagtgtgtg
960
gagaatagga ataacgtatg tgtagtcact aaacctctct gcatcagtaa gaacttgaat
1020
acagcaggga agtatatcag caaggtactt tctaaattca cacatccgta agtatctgcc
1080
agagttactc tctctgatat cagaaatatg gtcatctttc agcttgcctg ttgctaacag
1140
acctgttcac attgctccta ttttgcatct cgtggagcaa ctttgcttgg ctctcaacga
1200
tgaatttaga aagtaccttg ctgatatact tccctgctgt attcaagttc ttactgatgc
1260
agagaggttt agtgactaca catacgttat tcctattctc cacacacttg aagtttttgg
1320
tgggacatta gatgagcata tgcatctgct tcttcctgca cttattcggt tgtttaaatt
1380
ggatgcttca gtagaagtaa gacgcggtgc aatcaaaact ctcacaagat tgatacctcg
1440
tgtgcaggtc actggacaca tatcttctct tgtgcatcac ttgaagcttg tcttggacgg
1500
cccgggactc gaa
1513
<210> 107
<211> 1941
<212> ДНК
<213> Hordeum vulgare
<400> 107
atggccgcag ccacctccgc cgccgtcgca ttctcgggcg ccgccgccgc cgccgcggcc
60
ttacccaagc ccgccctcca tcctctcccg cgccaccagc ccgcctcgcg ccgcgcgctc
120
cccgcccgcg tcgtcaggtg ctgcgccgcg tcccccgccg ccaccacggc cgcgcctccc
180
cccacctctc tccggccgtg ggggccctcc gagccccgca agggcgccga catcctcgtc
240
gaggcgctcg agcgctgcgg catcgtcgac gtcttcgcct accccggcgg cgcgtccatg
300
gagatccacc aggcgctcac gcgctcgccc gtcatcacca accacctctt ccgccacgag
360
cagggggagg cgttcgcagc gtccgggtac gcacgcgcgt ccggccgcgt cggcgtctgc
420
gtcgccacct ccggccccgg ggccaccaac ctcgtctccg cgctcgccga cgctctcctc
480
gactccatcc ccatggtcgc catcacgggc caggtcccac gccgcatgat cggcacggac
540
gcgttccagg agacgcccat agtggaggtc acgcgctcca tcaccaagca caactacctg
600
gtccttgacg tggaggacat cccccgcgtc atccaggaag ccttcttcct cgcgtcctct
660
ggccgcccgg ggcctgtgct ggttgatatc cccaaggaca tccagcagca gatggccgtg
720
cctgtttggg acacgccgat gagtttgcca gggtacatcg cccgcctgcc caagccacca
780
tctactgaat cgcttgagca ggtcctgcgc ctggttggcg aggcacggcg cccgattctg
840
tatgttggtg gcggctgcgc tgcatctggc gaggagttgc gccgctttgt tgagctcact
900
ggaattccag ttacaactac tctgatgggc cttggcaact tccccagtga cgacccactg
960
tcactgcgca tgcttgggat gcatggtacc gtgtatgcaa attatgcagt agataaggct
1020
gacctgttgc ttgcatttgg tgtgcggttt gatgatcgcg tgactgggaa aattgaggct
1080
tttgcaagca ggtccaagat tgtgcacatt gacattgatc cagctgagat tggcaagaac
1140
aagcagccac atgtctccat ttgtgcagat gttaagcttg ctttacaggg gttgaatggt
1200
ctattaagtg gcagcaaagc acaacagggt ctagattttg gtccatggca caaggagttg
1260
gatcagcaga agagggagtt tcctctagga tacaagactt ttggtgaggc aatcccaccg
1320
cagtatgcta tccaggtact ggatgagctg acaaaagggg aggcgattat tgccacaggt
1380
gttgggcagc atcagatgtg ggcggctcag tattacactt acaagcggcc acgtcagtgg
1440
ctgtcttcgt ctggtttggg ggcaatggga tttgggttgc cagctgcagc tggcgcttct
1500
gtggccaacc caggtgtcac agttgttgac attgatgggg atggtagttt cctcatgaac
1560
attcaggagt tggcgttgat ccgtattgag aacctcccag tgaaggtgat gatattgaac
1620
aaccagcacc tgggaatggt ggtgcagtgg gaggataggt tttacaaggc caaccgggcg
1680
cacacatacc ttggcaaccc agaaaatgag agtgagatat atccagattt tgtgacgatt
1740
gctaaaggat tcaacgttcc ggcagttcgt gtgacaaaga agagtgaagt cagtgcagct
1800
atcaagaaga tgcttgagac cccagggccg tacctgctgg atatcattgt cccgcatcag
1860
gagcacgtgc tgcctatgat cccaagcggt ggtgctttca aggacatgat catggagggt
1920
gatggcagga cctcgtatta a
1941
<210> 108
<211> 1505
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая кпРНК, направленно
воздействующую на ген ALS ячменя (H. vulgare)
<400> 108
agatttaggt gacactatag aatggtggtg cagtgggagg ataggtttta caaggccaac
60
cgggcgcaca cataccttgg caacccagaa aatgagagtg agatatatcc agattttgtg
120
acgattgcta aaggattcaa cgttccggca gttcgtgtga caaagaagag tgaagtcagt
180
gcagctatca agaagatgct tgagacccca gggccgtacc tgctggatat cattgtcccg
240
catcaggagc acgtgctgcc tatgatccca aacggtggtg ctttcaagga catgatcatg
300
gagggtgatg gcaggacctc gtattaatct gaatttcgac ctacaagacc tacaagtgtg
360
acatgcgcaa tcagcatgat gcctgcgtgt tgtatcaact actaggggtt cgactgtgaa
420
ccatgcgttt ttctagttta cttgtttcat tcatataaga ggtcctgcca tcaccctcca
480
tgatcatgtc cttgaaagca ccaccgtttg ggatcatagg cagcacgtgc tcctgatgcg
540
ggacaatgat atccagcagg tacggccctg gggtctcaag catcttcttg atagctgcac
600
tgacttcact cttctttgtc acacgaactg ccggaacgtt gaatccttta gcaatcgtca
660
caaaatctgg atatatctca ctctcatttt ctgggttgcc aaggtatgtg tgcgcccggt
720
tggccttgta aaacctatcc tcccactgca ccaccattcc caggtgctgg ttgttcaata
780
tcatcacctt cactgggagg ttctcaatac ggatcaacgc caactcctga atgttcatga
840
ggaaactacc atccccatca atgtcaacaa ctgtgacacc tgggttggcc acagaagcgc
900
cagctgcagc tggcaaccca aatcccattg cccccaaacc agacgaagac agccactgac
960
gtggccgctt gtaagtgtaa tactgagccg cccacatctg atgctgccca acacctgtgg
1020
caataatcgc ctcccctttt gtcagctcat ccagtacctg aatggtctat taagtggcag
1080
caaagcacaa cagggtctag attttggtcc atggcacaag gagttggatc agcagaagag
1140
ggagtttcct ctaggataca agacttttgg tgaggcaatc ccaccgcagt atgctatcca
1200
ggtactggat gagctgacaa aaggggaggc gattattgcc acaggtgttg ggcagcatca
1260
gatgtgggcg gctcagtatt acacttacaa gcggccacgt cagtggctgt cttcgtctgg
1320
tttgggggca atgggatttg ggttgccagc tgcagctggc gcttctgtgg ccaacccagg
1380
tgtcacagtt gttgacattg atggggatgg tagtttcctc atgaacattc aggagttggc
1440
gttgatccgt attgagaacc tcccagtgaa ggtgatgata ttgaacaacc agcacctggc
1500
ccggg
1505
<210> 109
<211> 1824
<212> ДНК
<213> Hordeum vulgare
<400> 109
atgcagactc tgtcggcgca gcccctcgcc tcctcctctt cgatacagcg ccaccatggg
60
cgccgacgcg gccccggctc cgtccggttc gctccccgcg cggccgccgc ggctgccgcc
120
acgtccacca gcacggcccg ctcgccggcg tacgtctcgt cgccgtccac gaggaaggtg
180
cccgggtacg agcagtcgtc gccgcctgcc attgcctcgc cgcagaagca ggggagcagc
240
ggcggcgagg gcgagcagag cctcaacttc ttccagcgcg cggcggccgc ggcgctcgac
300
gcgttcgagg aggggttcat caacaatgtc ctggagcggc cccacgcgct gccgcgcacg
360
gccgacccgg ccgtgcagat cgccggcaac ttcgcccccg tcggcgagca gccccccgtg
420
cgcgccctca cggtctccgg ccgcatcccg cccttcatca acggcgtcta cgcccgcaac
480
ggcgccaacc cctgcttcga gcccacggcc ggccaccacc tcttcgacgg cgacggcatg
540
gtccacgcca tccgcatccg aaacggcgcc gccgagtcct acgcctgccg cttcaccgag
600
accgcccgcc tctcccagga gcgcgccgcg gggaggcccg tcttccctaa gaccatcggc
660
gagctccacg gccactctgg catcgcgagg ctggccctct tctacgcgcg cggcgcctgc
720
ggcctcgtcg acccgtccca cggcactggt gttgccaacg ccggcctcgt ctacttcaac
780
ggccgcctcc tcgccatgtc cgaggacgac ctcccgtacc aggtccgcgt caccgccggt
840
ggcgacctcg agaccgtcgg ccgctacgac ttcgacggcc agctcgactg cgccatgatc
900
gcgcacccca agctcgaccc tgtctccggc gagctcttcg cgctcagcta cgatgtcatc
960
aagaagccgt acctcaagta cttctacttc cacgccgacg gcaccaagtc cgccgacgtc
1020
gagatcgagc tcgaccagcc caccatgatc cacgacttcg ccatcaccga gaacttcgtc
1080
gtcgtgcccg accaccagat ggtgttcaag ctcgccgaga tgttccgcgg cggctcgccg
1140
gtgatgctcg acaaggagaa gacctcccgc ttcggcgtcc tcccaaagta cgccaaggac
1200
tcgtcggaga tgatgtgggt ggacgtgccg gactgcttct gtttccacct ctggaactcg
1260
tgggaggagc cggagacgga cgaggtggtg gtgatcggct cctgcacgac ccccgcagac
1320
tccatcttca acgacacgga cgaccacctc gagagcgtgc tcaccgagat ccggctcaac
1380
acgcgcaccg gcgagtccac gcggcgggcc atcctgccgc tggagagcca ggtgaacctc
1440
gaggtcggca tggtgaaccg caacatgctg ggccggaaga cgaggtacgc ctacctggcc
1500
gtggccgagc cgtggcccaa ggtgtccggg ttcgccaagg tggacctggt gaccggcgag
1560
ctgaccaagt tcgagtacgg cgagggccgg ttcggcggcg agccgtgctt cgtgcccatg
1620
gacggcgagc acgcgcgccc cggcgccgag gacgacggct acgtgctctc cttcgtgcgc
1680
gacgaggacg ccggcacatc cgagctcctg gtcgtcaacg ccgccgacat gcggctcgag
1740
gccaccgtgc agctgccgtc ccgggtcccc tatggcttcc acggcacatt catcggcgac
1800
gccgacctcg acgcccagca ctaa
1824
<210> 110
<211> 1779
<212> ДНК
<213> Hordeum vulgare
<400> 110
atgcagacac tcacagcgtc cagctcggtc tcctccatac agcggcaccg gccgcacccc
60
gcgggccgcc ggtccagctc ggtcaccttc tccgcccgcg ccgtcagctc cgcgccgcgc
120
gcgccggcac cgtcccggtt cgtgcgcggc gccgacgcgg cgcccgccaa gcccctcatt
180
gccgtcccca agccgcccgc cgtggagagg caggagaaga agctcaactt cttccagcgc
240
gccgcggtca cggcgctcga cgcgttcgag gaaggatttg tggccaacgt gctcgagcgc
300
ccgcacggcc tctccaggac ggtcgacccc gcggtgcaga tcgccggcaa cttcgcgcct
360
gtcggggaga cacctcctgt gcaggcgctg cccgtgaccg accgcatccc cccgttcatc
420
aacggcgtgt acgcccgcaa cggcgccaac ccgcacttcg accccgtcgc cgggcaccac
480
ctgttcgacg gcgacggcat ggtgcacgct ctgcgcatcc gcaacggcgt cgccgagacc
540
tacgcctccc gcttcaccga gacggagcgc ctgcagcagg agcgcgcgct ggggcgcccg
600
atgttcccca aggccattgg tgagctccat ggccactctg ggatcgcgcg ccttgctctg
660
ttctacgcgc gcgcggcctg cggcctcatc gacccctcgc gcggcaccgg cgtggccaac
720
gccggcctgg tctacttcaa cggccacctc ctcgccatgt ccgaggacga catcccgtac
780
cacgtccgcg tcaccgacga cggcgacctc cagaccgtcg gccgctacga cttcgacggg
840
cagctcgagt gccccatgat cgcgcacccc aaactcgacc ccgccaccgg ggagctccac
900
gcgctcagct acgacgtcat caagaagcct tacctgaagt acttctactt cgcggccgac
960
ggcaccaagt cggccgacgt cgagatcccg ctggaccagc ccaccatgat ccacgacttc
1020
gccatcaccg agaattacgt ggtcgtgccc gaccaccagg tggtgttcaa gctgcaggag
1080
atgctgcgcg gcggctcgcc cgtggtgctc gacaaggaga agacgtcccg cttcggcgtg
1140
ctgcccaagt gcgccgccga cgcgtcggag atggtgtggg tggacgtgcc ggactgcttc
1200
tgcttccacc tctggaacgc gtgggaggag gaggagaccg acgaggtggt ggtgatcggc
1260
tcctgcatga cccccgccga ctccatcttc aacgagtcgg acgagtgcct cgagagcgtg
1320
ctcacggaga tccgcctcaa cacccgcacc ggcgagtcca cgcggcgccc catcctggcg
1380
ctgtcagagc aggtgaacct ggaggtcggc atggtgaact ccaacctgct gggccgcaag
1440
acgcggtacg cctacctggc cgtggccgag ccgtggccca aggtgtccgg cttcgccaag
1500
gtcgacctgg ccacgggcga gctcaccaaa ttcgagtacg gcgagggccg gttcggcggc
1560
gagccctgct tcgtgcccat ggacccggcc acgtcccgcg gcgaggacga cgggtacatt
1620
ctcaccttcg tgcacgacga ggccgccggc acgtcggagc tgctggtggt caatgccgcc
1680
gacatgcggc tggaggcgac catccagctg ccgtcccgcg tgccatacgg gttccacggc
1740
accttcatca ccggcaagga gctcgaatcc caggcctga
1779
<210> 111
<211> 1500
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая конструкцию кпРНК, направленно
воздействующую на гены NCED1 ячменя Hordeum vulgare и пшеницы
Triticum aestivum
<400> 111
taatacgact cactataggg tcgacgaggc cgcaggcgcc gcgcgcgtag aagagggcca
60
gcctcgcgat gccagagtgg ccgtggagct cgccgatggt cttagggaag acgggcctcc
120
ccgcggcgcg ctcctgggag aggcgggcgg tctcggtgaa gcggcaggcg taggactcgg
180
cggcgccgtt tcggatgcgg atggcgtgga ccatgccgtc gccgtcgaag aggtggtggc
240
cggccgtggg ctcgaagcag gggttggcgc cgttgcgggc gtagacgccg ttgatgaagg
300
gcgggatgcg gccggagacc gtgagggcgc gcacgggggg ctgctcgccg acgggggcga
360
agttgccggc gatctgcacg gccgggtcgg ccgtgcgcgg cagcgcgtgg ggccgctcca
420
ggacattgtt gatgaacccc tcctcgaacg cgtcgagctc cggccgcatc ccgcccttca
480
tcaacggcgt ctacgcccgc aacggcgcca acccctgctt cgagcccacg gccggccacc
540
acctcttcga cggcgacggc atggtccacg ccatccgcat ccgaaacggc gccgccgagt
600
cctacgcctg ccgcttcacc gagaccgccc gcctctccca ggagcgcgcc gcggggaggc
660
ccgtcttccc taagaccatc ggcgagctcc acggccactc tggcatcgcg aggctggccc
720
tcttctacgc gcgcggcgcc tgcggcctcg tcgacccgta ccacggcact ggtgttgcca
780
acgccggcct cgtctacttc aacggccgcc tcctcgccat gtccgaggac gacctcccgt
840
accaggtccg cgtcaccgcc ggtggcgacc tcgagaccgt cggccgctac gacttcgacg
900
gccagctcga ctgcgccatg atcgcgcacc ccaagctcga ccctgtctcc ggcgagctct
960
tcgcgctcag ctacgatgtc atcaagaagc cgtacctcaa gtacttctac ttcacgcccg
1020
acggcaccaa gtccgccgac gtcgagatcg agctcgacga agcgggaggt cttctccttg
1080
tcgagcatca ccggcgagcc gccgcggaac atctcggcga gcttgaacac catctggtgg
1140
tcgggcacga cgacgaagtt ctcggtgatg gcgaagtcgt ggatcatggt gggctggtcg
1200
agctcgatct cgacgtcggc ggacttggtg ccgtcgggcg tgaagtagaa gtacttgagg
1260
tacggcttct tgatgacatc gtagctgagc gcgaagagct cgccggagac agggtcgagc
1320
ttggggtgcg cgatcatggc gcagtcgagc tggccgtcga agtcgtagcg gccgacggtc
1380
tcgaggtcgc caccggcggt gacgcggacc tggtacggga ggtcgtcctc ggacatggcg
1440
aggaggcggc cgttgaagta gacgaggccg gcgttggcaa caccagtgcc gtggtacgta
1500
<210> 112
<211> 1500
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая конструкцию кпРНК, направленно
воздействующую на гены NCED2 ячменя Hordeum vulgare и пшеницы
Triticum aestivum
<400> 112
taatacgact cactataggg ttggcgccgt tgcgggcgta cacgccgttg atgaacgggg
60
ggatgcggtc ggtcacgggc agcgcctgca caggaggtgt ctccccgaca ggcgcgaagt
120
tgccggcgat ctgcaccgcg gggtcgaccg tcctggagag gccgtgcggg cgctcgagca
180
cgttggccac aaatccttcc tcgaacgcgt cgagcgccgt gaccgcggcg cgctggaaga
240
agttgagctt cttctcctgc ctctccacgg cgggcggctt ggggacggca atgaggggct
300
tggcgggcgc cgcgtcggcg ccgcgcacga accgggacgg tgccggcgcg cgcggcgcgg
360
agctgacggc gcgggcggag aaggtgaccg agctggaccg gcggcccgcg gggtgcggcc
420
ggtgccgctg tatggaggag accgagctgg acgctgtcga cgcggcgccc gccaagcccc
480
tcattgccgt ccccaagccg cccgccgtgg agaggcagga gaagaagctc aacttcttcc
540
agcgcgccgc ggtcacggcg ctcgacgcgt tcgaggaagg atttgtggcc aacgtgctcg
600
agcgcccgca cggcctctcc aggacggtcg accccgcggt gcagatcgcc ggcaacttcg
660
cgcctgtcgg ggagacacct cctgtgcagg cgctgcccgt gaccgaccgc atccccccgt
720
tcatcaacgg cgtgtacgcc cgcaacggcg ccaacccgta cttcgacccc gtcgccgggc
780
accacctgtt cgacggcgac ggcatggtgc acgctctgcg catccgcaac ggcgtcgccg
840
agacctacgc ctcccgcttc accgagacgg agcgcctgca gcaggagcgc gcgctggggc
900
gcccgatgtt ccccaaggcc attggtgagc tccatggcca ctctgggatc gcgcgccttg
960
ctctgttcta cgcgcgcgcg gcctgcggcc tcatcgaccc ctcgcgcggc accggcgtgg
1020
ccaacgccgg cctggtctac ttcaacggcc acctcctccc cggtggcggg gtcgagtttg
1080
gggtgcgcga tcatggggca ctcgagctgc ccgtcgaagt cgtagcggcc gacggtctgg
1140
aggtcgccgt cgtcggtgac gcggacgtgg tacgggatgt cgtcctcgga catggcgagg
1200
aggtggccgt tgaagtagac caggccggcg ttggccacgc cggtgccgcg cgaggggtcg
1260
atgaggccgc aggccgcgcg cgcgtagaac agagcaaggc gcgcgatccc agagtggcca
1320
tggagctcac caatggcctt ggggaacatc gggcgcccca gcgcgcgctc ctgctgcagg
1380
cgctccgtct cggtgaagcg ggaggcgtag gtctcggcga cgccgttgcg gatgcgcaga
1440
gcgtgcacca tgccgtcgcc gtcgaacagg tggtgcccgg cgacggggtc gaagtacgta
1500
<210> 113
<211> 1521
<212> ДНК
<213> Hordeum vulgare
<400> 113
atggccttct tcctcctcct gtgcatcctc gtctctgtgg ccatcgtgtc ctacgcccac
60
cacgcaatcc ggcggaggcg ccagggctgc gctcatggcc gtcatgagca ggccgccctc
120
aagctgcccc ccggctccat gggcctgcct tacgtcggcg agaccctgca gctctactcc
180
caggacccca gcgtcttcct ctcctccaag cagaagcggt acggcgagat cttcaagacg
240
cacctcctgg ggtgcccgtg cgtgatgctg gcgagcccgg aggcggcgcg cttcgtgctg
300
gtgtcgcggg cccacctctt caagccgacg tacccgcgga gcaaggagcg cctcatcggc
360
ccgtcggcgc tcttcttcca ccagggcgac taccacctcc gcctccgccg gctcgtccag
420
ggcccgctcg gccccgaggc cctgcgcaag ctcgtgccgg acatcgaggc cgccgttcgc
480
tccacgctcg ccgcctgggc ggacggcgac gtcgccagca ctttccacgc catgaagagg
540
ctctcgttcg acgtcggcat cgtgacgatc ttcggcgggc ggctggacga gcggcggaag
600
gaggagctca ggcggaacta cgccgtcgtg gagaaaggct acaactcctt ccccaacagc
660
ttccccggga cgctatacta caaggcgatc caggcgaggc ggcggctgaa cggcgtgctg
720
agcgacgtcg tgcacgagcg tagggagcgg ggcgagcacg gcgacgacct cctcggctgc
780
ctcatgcggt cgcgggccgg cggcgacgac gccgacgacg agggcgcgct gctgacggac
840
gagcaggtcg ccgacaacgt catcggcgtg ctgttcgcgg cgcaggacac gacggccagc
900
gtgctcacct ggatcgtcaa gtacctccac gaccgcccga agctgctcga ggccgtcagg
960
gcggagcacg cggcgatcca cgaggccaac gacggcggga ggcggccgct gacatgggcg
1020
cagacgagga gcatgacgct gacgcacagg gtgattttgg agagcctaag gatggccagc
1080
atcatctcct tcacgttcag ggaggccgtg gccgacgtgg agtacaaagg gtttcttatc
1140
cccaaggggt ggaaggtgat gccgctcttc aggaacatcc atcacagccc ggactacttc
1200
caggatccac acaagttcga cccttcgcga ttcaaggtgg cgccgcggcc gaacaccttc
1260
acsccgttcg ggagcggggt gcacgcgtgc ccggggaacg agctggccaa gctcgagatg
1320
ctggtgctca tccaccacct ggtcaccggc tacaggtggg aggttgttgg atcgagcgac
1380
gacgtcgagt acagcccatt ccccgttccc cgccatggcc tgctcgccag ggtacggcga
1440
gatgacggcg tctgcgcggg taggaagggg tgcccgactg atgaagatga caactacgac
1500
gacgacgaag tgatagtgtg a
1521
<210> 114
<211> 1506
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая конструкцию кпРНК, направленно
воздействующую на гены ABA-OH-2 ячменя Hordeum vulgare и
пшеницы
Triticum aestivum
<400> 114
taatacgact cactataggg cggtcgtgga ggtacttgac gatccaggtg agcacgctgg
60
ccgtcgtgtc ctgcgccgcg aacagcacgc cgatgacgtt gtcggcgacc tgctcgtccg
120
tcagcagcgc gccctcgtcg tcggcgtcgt cgccgccggc ccgcgaccgc atgaggcagc
180
cgaggaggtc gtcgccgtgc tcgccccgct ccctacgctc gtgcacgacg tcgctcagca
240
cgccgttcag ccgccgcctc gcctggatcg ccttgtagta tagcgtcccg gggaagctgt
300
tggggaagga gttgtagcct ttctccacga cggcgtagtt ccgcctgagc tcctccttcc
360
gccgctcgtc cagccgcccg ccgaagatcg tcacgatgcc gacgtcgaac gagagcctct
420
tcatggcgtg gaaagtgctg gcgacgtcgc cgtccgccta caactccttc cccaacagct
480
tccccgggac gctatactac aaggcgatcc aggcgaggcg gcggctgaac ggcgtgctga
540
gcgacgtcgt gcacgagcgt agggagcggg gcgagcacgg cgacgacctc ctcggctgcc
600
tcatgcggtc gcgggccggc ggcgacgacg ccgacgacga gggcgcgctg ctgacggacg
660
agcaggtcgc cgacaacgtc atcggcgtgc tgttcgcggc gcaggacacg acggccagcg
720
tgctcacctg gatcgtcaag tacctccacg accgcccgta gctgctcgag gccgtcaggg
780
cggagcacgc ggcgatccac gaggccaacg acggcgggag gcggccgctg acatgggcgc
840
agacgaggag catgacgctg acgcacaggg tgattttgga gagcctaagg atggccagca
900
tcatctcctt cacgttcagg gaggccgtgg ccgacgtgga gtacaaaggg tttcttatcc
960
ccaaggggtg gaaggtgatg ccgctcttca ggaacatcca tcacagcccg gactacttcc
1020
aggatccaca caagttcgac ccttcgcgat tcaaggtcgc tcgatccaac aacctcccac
1080
ctgtagccgg tgaccaggtg gtggatgagc accagcatct cgagcttggc cagctcgttc
1140
cccgggcacg cgtgcacccc gctcccgaac ggagtgaagg tgttcggccg cggcgccacc
1200
ttgaatcgcg aagggtcgaa cttgtgtgga tcctggaagt agtccgggct gtgatggatg
1260
ttcctgaaga gcggcatcac cttccacccc ttggggataa gaaacccttt gtactccacg
1320
tcggccacgg cctccctgaa cgtgaaggag atgatgctgg ccatccttag gctctccaaa
1380
atcaccctgt gcgtcagcgt catgctcctc gtctgcgccc atgtcagcgg ccgcctcccg
1440
ccgtcgttgg cctcgtggat cgccgcgtgc tccgccctga cggcctcgag cagctacgta
1500
ggtacc
1506
<210> 115
<211> 3885
<212> ДНК
<213> Arabidopsis thaliana
<400> 115
atggaagctg aaattgtgaa tgtgagacct cagctagggt ttatccagag aatggttcct
60
gctctacttc ctgtcctttt ggtttctgtc ggatatattg atcccgggaa atgggttgca
120
aatatcgaag gaggtgctcg tttcgggtat gacttggtgg caattactct gcttttcaat
180
tttgccgcca tcttatgcca atatgttgca gctcgcataa gcgttgtgac tggtaaacac
240
ttggctcaga tctgcaatga agaatatgac aagtggacgt gcatgttctt gggcattcag
300
gcggagttct cagcaattct gctcgacctt accatggttg tgggagttgc gcatgcactt
360
aaccttttgt ttggggtgga gttatccact ggagtgtttt tggccgccat ggatgcgttt
420
ttatttcctg ttttcgcctc tttccttgaa aatggtatgg caaatacagt atccatttac
480
tctgcaggcc tggtattact tctctatgta tctggcgtct tgctgagtca gtctgagatc
540
ccactctcta tgaatggagt gttaactcgg ttaaatggag agagcgcatt cgcactgatg
600
ggtcttcttg gcgcaagcat cgtccctcac aatttttata tccattctta ttttgctggg
660
gaaagtacat cttcgtctga tgtcgacaag agcagcttgt gtcaagacca tttgttcgcc
720
atctttggtg tcttcagcgg actgtcactt gtaaattatg tattgatgaa tgcagcagct
780
aatgtgtttc acagtactgg ccttgtggta ctgacttttc acgatgcctt gtcactaatg
840
gagcaggtat ttatgagtcc gctcattcca gtggtctttt tgatgctctt gttcttctct
900
agtcaaatta ccgcactagc ttgggctttc ggtggagagg tcgtcctgca tgacttcctg
960
aagatagaaa tacccgcttg gcttcatcgt gctacaatca gaattcttgc agttgctcct
1020
gcgctttatt gtgtatggac atctggtgca gacggaatat accagttact tatattcacc
1080
caggtcttgg tggcaatgat gcttccttgc tcggtaatac cgcttttccg cattgcttcg
1140
tcgagacaaa tcatgggtgt ccataaaatc cctcaggttg gcgagttcct cgcacttaca
1200
acgtttttgg gatttctggg gttgaatgtt gtttttgttg ttgagatggt atttgggagc
1260
agtgactggg ctggtggttt gagatggaat accgtgatgg gcacctcgat tcagtacacc
1320
actctgcttg tatcgtcatg tgcatcctta tgcctgatac tctggctggc agccacgccg
1380
ctgaaatctg cgagtaacag agcggaagct caaatatgga acatggatgc tcaaaatgct
1440
ttatcttatc catctgttca agaagaggaa attgaaagaa cagaaacaag gaggaacgaa
1500
gacgaatcaa tagtgcggtt ggaaagcagg gtaaaggatc agttggatac tacgtctgtt
1560
actagctcgg tctatgattt gccagagaac attctaatga cggatcaaga aatccgttcg
1620
agccctccag aggaaagaga gttggatgta aagtactcta cctctcaagt tagtagtctt
1680
aaggaagact ctgatgtaaa ggaacagtct gtattgcagt caacagtggt taatgaggtc
1740
agtgataagg atctgattgt tgaaacaaag atggcgaaaa ttgaaccaat gagtcctgtg
1800
gagaagattg ttagcatgga gaataacagc aagtttattg aaaaggatgt tgaaggggtt
1860
tcatgggaaa cagaagaagc taccaaagct gctcctacaa gcaactttac tgtcggatct
1920
gatggtcctc cttcattccg cagcttaagt ggggaagggg gaagtgggac tggaagcctt
1980
tcacggttgc aaggtttggg acgtgctgcc cggagacact tatctgcgat ccttgatgaa
2040
ttttggggac atttatatga ttttcatggg caattggttg ctgaagccag ggcaaagaaa
2100
ctagatcagc tgtttggcac tgatcaaaag tcagcctctt ctatgaaagc agattcgttt
2160
ggaaaagaca ttagcagtgg atattgcatg tcaccaactg cgaagggaat ggattcacag
2220
atgacttcaa gtttatatga ttcactgaag cagcagagga caccgggaag tatcgattcg
2280
ttgtatggat tacaaagagg ttcgtcaccg tcaccgttgg tcaaccgtat gcagatgttg
2340
ggtgcatatg gtaacaccac taataataat aatgcttacg aattgagtga gagaagatac
2400
tctagcctgc gtgctccatc atcttcagag ggttgggaac accaacaacc agctacagtt
2460
cacggatacc agatgaagtc atatgtagac aatttggcaa aagaaaggct tgaagcctta
2520
caatcccgtg gagagatccc gacatcgaga tctatggcgc ttggtacatt gagctataca
2580
cagcaacttg ctttagcctt gaaacagaag tcccagaatg gtctaacccc tggaccagct
2640
cctgggtttg agaattttgc tgggtctaga agcatatcgc gacaatctga aagatcttat
2700
tacggtgttc catcttctgg caatactgat actgttggcg cagcagtagc caatgagaaa
2760
aaatatagta gcatgccaga tatctcagga ttgtctatgt ccgcaaggaa catgcattta
2820
ccaaacaaca agagtggata ctgggatccg tcaagtggag gaggagggta tggtgcgtct
2880
tatggtcggt taagcaatga atcatcgtta tattctaatt tggggtcacg ggtgggagta
2940
ccctcgactt atgatgacat ttctcaatca agaggaggct acagagatgc ctacagtttg
3000
ccacagagtg caacaacagg gaccggatcg ctttggtcca gacagccctt tgagcagttt
3060
ggtgtagcgg agaggaatgg tgctgttggt gaggagctca ggaatagatc gaatccgatc
3120
aatatagaca acaacgcttc ttctaatgtt gatgcagagg ctaagcttct tcagtcgttc
3180
aggcactgta ttctaaagct tattaaactt gaaggatccg agtggttgtt tggacaaagc
3240
gatggagttg atgaagaact gattgaccgg gtagctgcac gagagaagtt tatctatgaa
3300
gctgaagctc gagaaataaa ccaggtgggt cacatggggg agccactaat ttcatcggtt
3360
cctaactgtg gagatggttg cgtttggaga gctgatttga ttgtgagctt tggagtttgg
3420
tgcattcacc gtgtccttga cttgtctctc atggagagtc ggcctgagct ttggggaaag
3480
tacacttacg ttctcaaccg cctacaggga gtgattgatc cggcgttctc aaagctgcgg
3540
acaccaatga caccgtgctt ttgccttcag attccagcga gccaccagag agcgagtccg
3600
acttcagcta acggaatgtt acctccggct gcaaaaccgg ctaaaggcaa atgcacaacc
3660
gcagtcacac ttcttgatct aatcaaagac gttgaaatgg caatctcttg tagaaaaggc
3720
cgaaccggta cagctgcagg tgatgtggct ttcccaaagg ggaaagagaa tttggcttcg
3780
gttttgaagc ggtataaacg tcggttatcg aataaaccag taggtatgaa tcaggatgga
3840
cccggttcaa gaaaaaacgt gactgcgtac ggatcattgg gttga
3885
<210> 116
<211> 1080
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая конструкцию кпРНК, направленно
воздействующую на ген EIN2 A. thaliana
<400> 116
taatacgact cactataggg agctgcaaca tattggcata agatggcggc aaaattgaaa
60
agcagagtaa ttgccaccaa gtcatacccg aaacgagcac ctccttcgat atttgcaacc
120
catttcccgg gatcaatata tccgacagaa accaaaagga caggaagtag agcaggaacc
180
attctctgga taaaccctag ctgaggtctc acattcacaa tttcagcttc catcctaaat
240
ctatctgata atataattac tcagagtagg attcaaggta aactctacag tcgtggttca
300
ctaaaagcct cagttgagta aaattcacag atttgatcta aaacacctga atgtgagacc
360
tcagctaggg tttatccaga gaatggttcc tgctctactt cctgtccttt tggtttctgt
420
cggatatatt gatcccggga aatgggttgc aaatatcgaa ggaggtgctc gtttcgggta
480
tgacttggtg gcaattactc tgcttttcaa ttttgccgcc atcttatgcc aatatgttgc
540
agctcccaac agcgttgtga ctggtaaaca cttggctcag atctgcaatg aagaatatga
600
caagtggacg tgcatgttct tgggcattca ggcggagttc tcagcaattc tgctcgacct
660
taccatggtt gtgggagttg cgcatgcact taaccttttg tttggggtgg agttatccac
720
tggagtgttt ttggccgcca tggatgcgag tgggatctca gactgactca gcaagacgcc
780
agatacatag agaagtaata ccaggcctgc agagtaaatg gatactgtat ttgccatacc
840
attttcaagg aaagaggcga aaacaggaaa taaaaacgca tccatggcgg ccaaaaacac
900
tccagtggat aactccaccc caaacaaaag gttaagtgca tgcgcaactc ccacaaccat
960
ggtaaggtcg agcagaattg ctgagaactc cgcctgaatg cccaagaaca tgcacgtcca
1020
cttgtcatat tcttcattgc agatctgagc caagtgttta ccagtcacaa cgctgttgac
1080
<210> 117
<211> 1188
<212> ДНК
<213> Arabidopsis thaliana
<400> 117
atggtgatgg ctggtgcttc ttctttggat gagatcagac aggctcagag agctgatgga
60
cctgcaggca tcttggctat tggcactgct aaccctgaga accatgtgct tcaggcggag
120
tatcctgact actacttccg catcaccaac agtgaacaca tgaccgacct caaggagaag
180
ttcaagcgca tgtgcgacaa gtcgacaatt cggaaacgtc acatgcatct gacggaggaa
240
ttcctcaagg aaaacccaca catgtgtgct tacatggctc cttctctgga caccagacag
300
gacatcgtgg tggtcgaagt ccctaagcta ggcaaagaag cggcagtgaa ggccatcaag
360
gagtggggcc agcccaagtc aaagatcact catgtcgtct tctgcactac ctccggcgtc
420
gacatgcctg gtgctgacta ccagctcacc aagcttcttg gtctccgtcc ttccgtcaag
480
cgtctcatga tgtaccagca aggttgcttc gccggcggta ctgtcctccg tatcgctaag
540
gatctcgccg agaacaatcg tggagcacgt gtcctcgttg tctgctctga gatcacagcc
600
gttaccttcc gtggtccctc tgacacccac cttgactccc tcgtcggtca ggctcttttc
660
agtgatggcg ccgccgcact cattgtgggg tcggaccctg acacatctgt cggagagaaa
720
cccatctttg agatggtgtc tgccgctcag accatccttc cagactctga tggtgccata
780
gacggacatt tgagggaagt tggtctcacc ttccatctcc tcaaggatgt tcccggcctc
840
atctccaaga acattgtgaa gagtctagac gaagcgttta aacctttggg gataagtgac
900
tggaactccc tcttctggat agcccaccct ggaggtccag cgatcctaga ccaggtggag
960
ataaagctag gactaaagga agagaagatg agggcgacac gtcacgtgtt gagcgagtat
1020
ggaaacatgt cgagcgcgtg cgttctcttc atactagacg agatgaggag gaagtcagct
1080
aaggatggtg tggccacgac aggagaaggg ttggagtggg gtgtcttgtt tggtttcgga
1140
ccaggtctca ctgttgagac agtcgtcttg cacagcgttc ctctctaa
1188
<210> 118
<211> 1080
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая конструкцию кпРНК, направленно
воздействующую на ген CHS A. thaliana
<400> 118
taatacgact cactataggg gccacaccat ccttagctga cttcctcctc atctcgtcta
60
gtatgaagag aacgcacgcg ctcgacatgt ttccatactc gctcaacacg tgacgtgtcg
120
ccctcatctt ctcttccttt agtcctagct ttatctccac ctggtctagg atcgctggac
180
ctccagggtg ggctatccag aagagggagt tccagtcact tatccccaaa ggtttaaacg
240
cttcgtctag actcttcaca atgttcttgg agatgaggcc gggaacatcc ttgaggagat
300
ggaaggtgag accaacttcc ctcaaatgtc cgtctatggc accatcagac tggaactccc
360
tcttctggat agcccaccct ggaggtccag cgatcctaga ccaggtggag ataaagctag
420
gactaaagga agagaagatg agggcgacac gtcacgtgtt gagcgagtat ggaaacatgt
480
cgagcgcgtg cgttctcttc atactagacg agatgaggag gaagtcagct aaggatggtg
540
tggccccgac aggagaaggg ttggagtggg gtgtcttgtt tggtttcgga ccaggtctca
600
ctgttgagac agtcgtcttg cacagcgttc ctctctaaac agaacgcttg ccttctatct
660
gcctacctac ctacgcaaaa ctttaatcct gtcttatgtt ttatataata taatcattat
720
atgtttacgc aataattaag gaagaatgac atttccaaac aaagatttga tgtcattcaa
780
gacccataga tttaatattg taaaaagaca caaaaaagag agtacaaaaa cagtcgaata
840
gacctgtcca gcacatatca catatcacat caaatgcatt cttccttaat tattgcgtaa
900
acatataatg attatattat ataaaacata agacaggatt aaagttttgc gtaggtaggt
960
aggcagatag aaggcaagcg ttctgtttag agaggaacgc tgtgcaagac gactgtctca
1020
acagtgagac ctggtccgaa accaaacaag acaccccact ccaacccttc tcctgtcaac
1080
<210> 119
<211> 3456
<212> ДНК
<213> Lupinus angustifolius
<400> 119
atgttgactc ttcaacccac acatgagtca agtagtcaat accctcctca tacacttata
60
gctgagacct gtcactttga ttatctgtac tatactaatc aaagttctct aattatgtca
120
cttggagaat catccctgca atggaaatac catgttttct tgagttttag gggaggtgac
180
acccgcttaa gcttcactaa tcacttatat gctgcgttgg tgcgaaaagg aatcattact
240
ttccgagatg acaaacaact tcacaaagga gatgccattt ctcaacatct gcatcaatca
300
atccaacagt ctctagctgc cattgttgtt atctcggaga actatgcttc ttccacttgg
360
tgtttggatg agctaaaact aattcttgaa tcgagaatag atgtttttcc agtcttttat
420
ggtgtcactc cttctgatgt tcgataccag aaaaatagtt ttgctgaggc tttcaataaa
480
catgttgtaa gatttgaaca agatgaagag aaagtgcaaa aatggagaga ttgcttgaaa
540
gaagttgctg atttttctgg atgggagtcc aaggacatgg ctgaagcaga actcattgaa
600
gatgttattg aaaaggtatg gataaaacta caaccaaaat tgccatccta caatgaagga
660
gtggttggat ttgattcaag ggtgaagaaa atgatttcac ttttaagcat aggatcacaa
720
gatattcggt ttatcgggat atggggtatg gctggaactg gaaaaacaat tcttgctaga
780
gtaatctacg aaacaataag tagccaattt gagattaaat gtttccttct taatgttaga
840
gaggtttctc aaacatctga tggattggtt tccttacaaa gaaaacttct ttctaccctt
900
aagataagca acctagaaat tgatgatttg tatgatggaa agaagaaaat tatgaacctt
960
ttgtgcaaca aaagtgttct tcttgtcctt gatgacatta gtcatttaag tcagctagag
1020
aatttggcta aaactaaagg ttggtttggt ccatgcagca gagtgataat aacaaccaaa
1080
gatatgcact tactagtatc acatggtgcg tgtgagaagt atgagatgag aatcttaaat
1140
gaaagttctt cctttcaact cttcagccag aaagcattca gaagagataa acctccagag
1200
ggttatttag aaataactaa aagtatggtc aaatatgctg gaggtcttcc tttggcactt
1260
aaagtgttgg gttcttttgt ttgtggaaga agtctcagtc agtggaagga tgctttggat
1320
aagataaaac aagttctgcc gaaagacatt ttgaacacac taataatagg ttatgatgga
1380
ctagaagatg cagaaaagac tttgttttta gatattgctt tcttctttac aggacggtcg
1440
aaaattgaag tgatacaggt attggcagat tgtggcctta atccaacaat tggaataagt
1500
cttcttattg aaagatctct agtaagttgt tgtggaggaa ttttggaaat gcatgattta
1560
cttcaagaaa tgggtagaaa tattgtatat caagaatctc cggatgatgc aagcagacgc
1620
agtaggttat gctctttaga agatattaac cgagtattca gaaaaaacaa gggaaccaat
1680
atcattcaag gaatagttct gaaatcaagt gacccatgtg aagcatattg gcatcctgaa
1740
gccttctcaa aaatggataa tcttagagta ctcatcattt tgtgtgattt gcaccttccc
1800
ctcggcctca aatgtctctc tagttcatta aaacttcttg aatggaaggg atatcctttg
1860
gaatatctac catttggcct gcaactgcta gaacttgttc acttgaaaat gcattgcagc
1920
aaacttaaac aactttggaa tggaactcaa attttcagag agctaaaatc aattgatctc
1980
agtgattcca gagatctaat tcaaactcca gatatttctg aggttccatg tcttgagagt
2040
ttagttttga aaggttgtaa aaaccttgtt gaggttcatc aatctgttgc aaagcacaag
2100
aatgttgcta tactagacct ggaaggttgc atcagtctta agaccctgcc aagaaaattg
2160
gagatgaatg ctttggaaaa gttcattctc tccggctgct cacaaattaa aaaccttccc
2220
gaatttgggg agagtatgga atgtctatct atgcttaatt taagagattg cacaagtctt
2280
gtttctcttc cacagagtgt tcgaaacatg aaatccttta gagatctcaa tatccatggt
2340
tgctcaaaat tgtttaagct gacaaacaat tcaaatgaaa ataatgtcgt ggaagaaatt
2400
gatgagactg aaacaggtag gagagaagtg cattcatcat ggagcttttc tctccttact
2460
gagaaagtgt ttgatttcgt aaagtatcca gttagcatgg actcgaagtt gccttctctc
2520
tcaagtttcc ctcggttgaa gaaattagat atgggcaact gtaatctcag tgatggacca
2580
attatagatc atattggaca tttaacatca ctggaagtgt tatatttagc tgggaacaac
2640
tttgttgacc ttacagcaag cattggtaac ctttctcggc tacaacgcct tggtttatat
2700
aaatgccgaa gacttaggac attgcctgag cttccaccca gtgtatgcca gttacttatg
2760
aacgactgca ctcaactgga acctatgtta tttgacacac aaataatttt gaaaatattt
2820
gaggcaaata gatggagcct gacacgcgaa ttgtggttcc tgattccagg gagtgaaatc
2880
ccagcatggt ttgagcatca agattatttt agcctgaaac caagtttagc gcctttcgat
2940
tatcacgagg agtatgcttt tattgtttca acaatagtaa acatccctga ctattgcctt
3000
tcaagtgatt ggataggaat tattgtatgc tttttactgg aaagtggttt aaaggcagac
3060
ctacacagac atattcgtag aagtccggtc acgatcggat ggtcttttaa agatcccgat
3120
gcagaaacgg tttacccctt acgcttcact aaacgtcgtt ggacacattt caaaggcaat
3180
cacctattga ttactacttt tggaagtgat catagaatat acaagcacta cttaacttgt
3240
ggcaaaagca aagtgcaatt gatattttgt ggtgagaata tttgcaagtg cgggaagcta
3300
aagctgaaaa actgtgggat ccgtgtgatt tgtaaggaag atggtgtatc gcgtagaggc
3360
gaggaaacga gtgaagttga ggtgccttcc acttcagttg aatctgatgt tcacaaacaa
3420
tcacgaataa ctgaaattac agatgaatat gaataa
3456
<210> 120
<211> 1280
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая конструкцию кпРНК, направленно
воздействующую на N-подобный ген L. angustifolius
<400> 120
taatacgact cactataggg tatcgaacat cagaaggagt gacaccataa aagactggaa
60
aaacatctat tctcgattca agaattagtt ttagctcatc caaacaccaa gtggaagagt
120
atttccattg cagggatgat tctccaagtg acataattag agaactttga ttagtatagt
180
acagataatc aaagtgacag gtctcagcta taagtgtatg aggagggtat tgactacttg
240
actcatgtgt gggttgaaga gtcaacatat agcttcacgg atcccacagt ttttcagctt
300
tagcttcccg cacttgcaaa tattctcacc acaaaatatc aattgcactt tgcttttgcc
360
acaagttaag tagtgcttgt atattctatg atcaccctgt tgactcttca acccacacat
420
gagtcaagta gtcaataccc tcctcataca cttatagctg agacctgtca ctttgattat
480
ctgtactata ctaatcaaag ttctctaatt atgtcacttg gagaatcatc cctgcaatgg
540
aaatactctt ccacttggtg tttggatgag ctaaaactaa ttcttgaatc gagaatagat
600
gtttttccag tcttttatgg tgtcactcct tctgatgttc gatacccgta gttttgctga
660
ggctttcaat aaacatgttg taagatttga acaagatgaa gagaaagtgc agtttgagca
720
tcaagattat tttagcctga aaccaagttt agcgcctttc gattatcacg aggagtatgc
780
ttttattgtt tcaacaatag taaacatccc tgactattgc ctttcaagtg attggatagg
840
aattattgta tgctttttac tggaaagtgg tttaaaggca gacctacaca gacatattcc
900
aaaagtagta atcaataggt gattgccttt gaaatgtgtc caacgacgtt tagtgaagcg
960
taaggggtaa accgtttctg catcgggatc tttaaaagac catccgatcg tgaccggact
1020
tctacgaata tgtctgtgta ggtctgcctt taaaccactt tccagtaaaa agcatacaat
1080
aattcctatc caatcacttg aaaggcaata gtcagggatg tttactattg ttgaaacaat
1140
aaaagcatac tcctcgtgat aatcgaaagg cgctaaactt ggtttcaggc taaaataatc
1200
ttgatgctca aactgcactt tctcttcatc ttgttcaaat cttacaacat gtttattgaa
1260
agcctcagca aaactacgta
1280
<210> 121
<211> 1527
<212> ДНК
<213> Vitis pseudoreticulata
<400> 121
atggctggcg acgaggagac gacgacgacg gcagcaacac ttgaaacaac gtccacttgg
60
gctgttgcct ctgtttgctt tattttgatt gcactctcca tacttattga gcatgccctc
120
catctcttag ccaagtactt caacaagaag cggaggaggt ctctcattca tgctcttaac
180
aacgtcaaat cggagttgat gctcttgggg ttcgtctctt tgttgctgac tgtgtgccaa
240
aagtatattg cgaagatttg tatcccaagg agcgtaggtg aaacttttct tccctgcaag
300
accttgacag aaagtgattc agaagaagaa accaaatgcg aagagcaggg aaagatgtct
360
ttgctgtcta gacaaggcgt ggaggaacta caatacttaa ttttcgtgct ggccttcttc
420
cattccctct actgcgtcct cacattcggt cttgggatgg ccaagatgaa gaaatgggag
480
tcctgggagg cagaaacaag aacactggaa tatcagttta caaatgatcc acggaggttc
540
aggctcatcc atcagacatc atttggaaag caacatctga gatattggag tgagcatcag
600
atacttcgtt ggccggcttg ttttattcgg cagttctatc catccgtctc caaagtggat
660
tacttgactc ttagacatgg gttcattatg gcccattttg cagaaggaag caactatgac
720
ttccaaaagt atataaaaag agctttggaa aaagactttg gagtggtggt gggaggaagt
780
ttctgggttt ggagtttctc catgcttttt gtgttcttca atgctcaagt attttacaac
840
tatttatggc taccctttat tccattggtg atgctgttgt tggttggaac aaagctacag
900
ggcattataa ctaagatgtg cttagatagc catgataaag ctctcgttgt tagaggaact
960
ttgcttgtca ggcccagtga tcacttcttc tggtttggaa aaccggaatt gctcctacat
1020
cttatgcact ttatattgtt tcagaactct tttcaactgg cgttctttac atggacttgg
1080
tacaaatttg gattcagatc atgcttccat gatacaactg aggatatcgt cataaggctt
1140
gtcatgggtg tgttagtaca actcctttgt ggctacgtga cactgcctct gtatgccctg
1200
gtcacgcaga tggggacatc aatgaggaca attgtcttta ctgagggagt cgttgaaggt
1260
ctgaacagat ggagaaggaa agccaagaaa aacatagcac gcaggaacaa ccactcagct
1320
cgtccctccc tggatgcttc actcgacaat tcaccttctt ttaacactct ggatacttct
1380
ttctctgtag acctcgatca gccatcatca gatgctggtt atttgactgt tgaaatatca
1440
gatgaagaga cggtcgctac taaacggcca gaaccgcgtc agaagttggg atcttttgag
1500
ggtttcgact cgtgcaaaac atcataa
1527
<210> 122
<211> 1480
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая первую конструкцию кпРНК, направленно
воздействующую на ген MLO Vitis
<400> 122
taatacgact cactataggg tagccataaa tagttgtaaa atacttgagc attgaagaac
60
acaaaaagca tggagaaact ccaaacccag aaacttcctc ccaccaccac tccaaagtct
120
ttttccaaag ctctttttat atacttttgg aagtcatagt tgcttccttc tgcaaaatgg
180
gccataatga acccatgtct aagagtcaag taatccactt tggagacgga tggatagaac
240
tgctgaataa aacaaaccgg ccaacgaagt atccgatgct cactccaata tctcagatgt
300
tgctttccaa atgatgtctg atggatgagc gtgaacctcc gtggatcatt tgtaaactga
360
tattccagtg ttcttgtttc tgcctcccag gactcccatt tcttcatctt ggccatccca
420
agaccgaatg tgaggacgca gtagagcaca tcatttggaa agcaacatct gagatattgg
480
agtgagcatc ggatacttcg ttggccggtt tgttttattc agcagttcta tccatccgtc
540
tccaaagtgg attacttgac tcttagacat gggttcatta tggcccattt tgcagaagga
600
agcaactatg acttccaaaa gtatataaaa agagctttgg aaaaagactt tggagtggtg
660
gtgggaggaa gtttctgggt ttggagtttc tccatgcttt ttgtgttctt caatgctcaa
720
gtattttaca actatttatg gctacccgta attccattgg tgatgctgtt gttggttgga
780
acaaagctac agggcattat aactaagatg tgcctagata gccatgataa agctctcgtt
840
gttagaggaa ctttgcttgt caggcccagt gatcacttct tctggtttgg aaaaccggaa
900
ttgctcctac atcttatgca ctttatattg tttcagaact cttttcaact ggcgttcttt
960
acatggactt ggtacaaatt tggattcaga tcatgcttcc atgatacaac tgaggatatc
1020
gtcataaggc ttgtcatggg tgtgttatgg ctttccttct ccatctgttc agaccttcaa
1080
cgactccctc agtaaagaca attgtcctca ttgatgtccc catctgcgtg accagggcat
1140
acagaggcag tgtcacgtag ccacaaagga gttgtactaa cacacccatg acaagcctta
1200
tgacgatatc ctcagttgta tcatggaagc atgatctgaa tccaaatttg taccaagtcc
1260
atgtaaagaa cgccagttga aaagagttct gaaacaatat aaagtgcata agatgtagga
1320
gcaattccgg ttttccaaac cagaagaagt gatcactggg cctgacaagc aaagttcctc
1380
taacaacgag agctttatca tggctatcta ggcacatctt agttataatg ccctgtagct
1440
ttgttccaac caacaacagc atcaccaatg gaattacgta
1480
<210> 123
<211> 894
<212> ДНК
<213> Myzus persicae
<400> 123
atgttcaaac acttgtgcaa taccgtttca caaagtataa aacctagtag ttttttatca
60
aaagtttgtt caaacaaata tctcgtcgtg ccgtaccgga tagcgatttt taacaacatg
120
ggaagttaca aattgtacct ggccgtcatg gcaatagctg tcatagctgc agttcaggaa
180
attagttgca aggttcagac ttccgaacag gacgatgatc aggaaggata ttacgatgat
240
gagggaggag tgaacgataa tcagggagaa gagaacgata atcagggaga agagaacgat
300
aatcagggag aagagaacga taatcaggga gaagagaagg aagaagtttc cgaaccagag
360
atggagcacc atcagtgcga agaatacaaa tcgaagatct ggaacgatgc atttagcaac
420
ccgaaggcta tgaacctgat gaaactgacg tttaatacag ctaaggaatt gggctccaac
480
gaagtgtgct cggacacgac ccgggcctta tttaacttcg tcgatgtgat ggccaccagc
540
ccgtacgccc acttctcgct aggtatgttt aacaagatgg tggcgtttat tttgagggag
600
gtggacacga catcggacaa atttaaagag acgaagcagg tggtcgaccg tatctcgaaa
660
actccagaga tccgtgacta tatcaggaac tcggccgcca agaccgtcga cttgctcaag
720
gaacccaaga ttagagcacg actgttcaga gtgatgaaag ccttcgagag tctgataaaa
780
ccaaacgaaa acgaagcatt aatcaaacag aagattaagg ggttaaccaa tgctcccgtc
840
aagttagcca agggtgccat gaaaacggtt ggacgtttct ttagacattt ttaa
894
<210> 124
<211> 960
<212> ДНК
<213> Myzus persicae
<400> 124
atgactgaga caatgcaact ccgtggtacc cttcgtgggc ataatggttg ggttacgcag
60
atcgccacca atccgatcca cactgacatg attctgtctt gttcacgaga caagaccttg
120
attgtttggg atctgacacg tgatgagctc aactatggta tccccaagaa acgtttgtac
180
ggacattcgc acttcgtcag cgacgtcgtt ctttcatcag atggtaacta cgctctttcc
240
ggttcttggg ataagactct tcgtctgtgg gatttggctg ctggacgtac cactcgtcgt
300
tttgaagacc acaccaagga tgtattgagc gttgccttct ctgctgacaa ccgtcaaatc
360
gtttctggaa gtcgggacaa gactatcaag ttgtggaata ctttggctga gtgcaaatac
420
actattcagg atgatggaca tagcgattgg gtatcatgtg tacggttctc tcctaatatc
480
cataacccaa tcattgtgag tgctggttgg gacaaggttg tcaaggtatg gaacttaact
540
aactgccgca tcaagaccaa ccattatgga cacactggat accttaacac cgttactgtt
600
tcacctgatg gttctttgtg tgcttcagga ggaaaagatt gcaaagctat gttatgggat
660
cttaatgacg gcaaacactt gcacacactg gaccataacg atatcattga agctttgtgc
720
tttagcccca accgttactg gttgtgcgct gcatttggac catcaatcaa aatttgggat
780
ttggaaagca aagaaatggt tgaggaactt cgcccagaag ttgtatctca atcacagaat
840
agcaataccg aaccacccag atgtctgtca cttgcatggt caactgatgg acaaacattg
900
tttgctggat actcagacaa taacattaga gtttggcaag tgtctgtcag tgctcgttaa
960
<210> 125
<211> 1401
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная конструкция, кодирующая кпРНК, направленно
воздействующую
на ген C002 M.persicae
<400> 125
gaattctaat acgactcact atagggtgct ccatctctgg ttcggaaact tcttccttct
60
cttctccctg attatcgttc tcttctccct gattatcgtt ctcttctccc tgattatcgt
120
tctcttctcc ctgattatcg ttcactcctc cctcatcatc gtaatatcct tcctgatcat
180
cgtcctgttc ggaagtctga accttgcaac taatttcctg aactgcagct atgacagcta
240
ttgccatgac ggccaggtac aatttgtaac ttcccatgtt gttaaaaatc gctatccggt
300
acggcacgac gagatatttg tttgaacaaa cttttgataa aaaactacta ggttttatac
360
tttgtgaaac ggtattgcac aagtgtttga acataagaga gttggaagtt acaaattgta
420
cctggccgtc atggcaatag ctgtcatagc tgcagttcag gaaattagtt gcaaggttca
480
gacttccgaa caggacgatg atcaggaagg atattacgat gatgagggag gagtgaacga
540
taatcaggga gaagagaacg ataatcaggg agaagagaac gataatcagg gagaagagaa
600
cgataatcag ggagaagaga aggaagaagt ttccgaacca gagatggagc acccaacagt
660
gcgaagaata caaatcgaag atctggaacg atgcatttag caacccgaag gctatgaacc
720
tgatgaaact gacgtttaat acagctaagg aattgggctc caacgaagtg tgctcggaca
780
cgacccgggc cttatttaac ttcgtcgatg tgatggccac cagcccgtac gcccacttct
840
cgctaggtat gtttaacaag atggtggcgt ttattttgag ggaggtggac acgacatcgg
900
acaatctgaa cagtcgtgct ctaatcttgg gttccttgag caagtggacg gtcttggcgg
960
ccgagttcct gatatagtca cggatctctg gagttttcga gatacgggcg accacctgct
1020
tcgtctcttt aaatttgtcc gatgtcgtgt ccacctccct caaaataaac gccaccatct
1080
tgttaaacat acctagcgag aagtgggcgt acgggctggt ggccatcaca tcgacgaagt
1140
taaataaggc ccgggtcgtg tccgagcaca cttcgttgga gcccaattcc ttagctgtat
1200
taaacgtcag tttcatcagg ttcatagcct tcgggttgct aaatgcatcg ttccagatct
1260
tcgatttgta ttcttcgcac tgttaacaag cttagcatat ccatgatatc tgttagtttt
1320
tttcctgaaa gagcggccgc cctagcataa ccccgcgggg cctcttcggg ggtctcgcgg
1380
ggttttttgc tgaaaggatc c
1401
<210> 126
<211> 1401
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная конструкция, кодирующая кпРНК, направленно
воздействующую
на ген Rack-1 M.persicae
<400> 126
gaattctaat acgactcact atagggttat ggatattagg agagaaccgt acacatgata
60
cccaatcgct atgtccatca tcctgaatag tgtatttgca ctcagccaaa gtattccaca
120
acttgatagt cttgtcccga cttccagaaa cgatttgacg gttgtcagca gagaaggcaa
180
cgctcaatac atccttggtg tggtcttcaa aacgacgagt ggtacgtcca gcagccaaat
240
cccacagacg aagagtctta tcccaagaac cggaaagagc gtagttacca tctgatgaaa
300
gaacgacgtc gctgacgaag tgcgaatgtc cgtacaaacg tttcttgggg ataccatagt
360
tgagctcatc acgtgtcaga tcccaaacaa tcaaggtctt gtcccggttc ttgggataag
420
actcttcgtc tgtgggattt ggctgctgga cgtaccactc gtcgttttga agaccacacc
480
aaggatgtat tgagcgttgc cttctctgct gacaaccgtc aaatcgtttc tggaagtcgg
540
gacaagacta tcaagttgtg gaatactttg gctgagtgca aatacactat tcaggatgat
600
ggacatagcg attgggtatc atgtgtacgg ttctctccta atatccataa cccaaccatt
660
gtgagtgctg gttgggacaa ggttgtcaag gtatggaact taactaactg ccgcatcaag
720
accaaccatt atggacacac tggatacctt aacaccgtta ctgtttcacc tgatggttct
780
ttgtgtgctt caggaggaaa agattgcaaa gctatgttat gggatcttaa tgacggcaaa
840
cacttgcaca cactggacca taacgatatc attgaagctt tgtgctttag ccccaaccgt
900
tacacagaca tctgggtggt tcggtattgc tattctgtga ttgagataca acttctgggc
960
gaagttcctc aaccatttct ttgctttcca aatcccaaat tttgattgat ggtccaaatg
1020
cagcgcacaa ccagtaacgg ttggggctaa agcacaaagc ttcaatgata tcgttatggt
1080
ccagtgtgtg caagtgtttg ccgtcattaa gatcccataa catagctttg caatcttttc
1140
ctcctgaagc acacaaagaa ccatcaggtg aaacagtaac ggtgttaagg tatccagtgt
1200
gtccataatg gttggtcttg atgcggcagt tagttaagtt ccataccttg acaaccttgt
1260
cccaaccagc actcacaatg gttaacccgg gtagcatatc catgatatct gttagttttt
1320
ttcctgaaag agcggccgcc ctagcataac cccgcggggc ctcttcgggg gtctcgcggg
1380
gttttttgct gaaaggatcc c
1401
<210> 127
<211> 2396
<212> ДНК
<213> Helicoverpa armigera
<400> 127
agacattgat tagtgagctc caaactccgt acgtacgttc ttagtttagt ttgttcgttc
60
gtattgtcgc agtcacatcg ctccggtgcc cgcttcgaca tttcccgcca aaagtgacgt
120
aacatatccg tgatctgtgt gaatatgtca gtgacttttt taaattaatt ttttaatagc
180
aaaattgtga tcgaaggaat ttttacaaga tgacggctgg gaatgaagag catgagcctc
240
taattacatc gtctgtcgac aatcagcgtg tggcctacag taattcacca ccggatgacc
300
gcacaccaga atcttcttcc ccacgcggca gtggcggaga agtaacgcta gccataccat
360
cacaccgcaa ctatggagcc atcggaggcg tggagaaggt cacatacacc tgggcagaca
420
tcaatgcctt tgctactgaa tccaggtcta ggtcccgaag gatttggaac ttctggaagc
480
cctccgccag tggcatgttc cagcaaagga aacagttgtt gaggaatgta aatggagccg
540
cctacccagg cgaactgctc gccatcatgg gatcctccgg tgccgggaag accacactcc
600
tcaacactct gaccttccgc actccaagcg gggtgctgtc cagtggcact cgagcactga
660
acggccagcc tgctacccct gaggcgttat cagcactgtc tgcgtatgtt cagcagcagg
720
atctgttcat tggcacgctg actgtgaagg agcatttagt attccaggct atggtgcgga
780
tggaccgaca tataccgtat gcgcagcgca tgaggagagt tcaagaggtt attactgagt
840
tggcgctaac aaaatgccag aacacagtga taggcatccc tgggcggctg aagggtatct
900
ccggcgggga gatgaagagg ctgtccttcg ccagcgaggt gctcacggat ccaccgctca
960
tgttctgcga tgaacccacc tctggactcg attcttttat ggcgcagaat gttatacagg
1020
tactgaaagg tctcgcacaa aaaggcaaga cagtcgtatg cacgatccac cagccgtctt
1080
cggagctgta cgcgatgttc gataagctgc tcatcatggc agacgggaag gtcgccttcc
1140
tcggctcccc tgatcaggct aatgatttct ttaaagacct aggagcagcg tgtcctccta
1200
actacaaccc agcggaccac ttcatccaac tcctggcggg agtgccgggc agggaggaga
1260
ccacgcgcac cactatcgat actgtctgca cggcattcgc gcgctctgag gtcggctgca
1320
agattgctgc agaagctgaa aatgcactct actttgagcg caagatatcg cagggctggg
1380
cggacccggc gtggtctgaa gccacggcta tccgcgcgcg ccgctcgccg tacaaggcgt
1440
cgtggtgcgc gcagttccgc gcggtgctgt ggcgctcgtg gctgtccgtc actaaggagc
1500
ccatgctcat caaagtgcgc ttcctacaga ctattatggt atcgatcctg atcggcgtga
1560
tctacttcgg gcagcacctg gaccaggacg gcgtgatgaa catcaacggc gccatcttca
1620
tgttcctcac caacatgacc ttccagaaca tcttcgctgt tattaacgta ttctgctcag
1680
aactgccaat attcatacga gaacaccact ccgggatgta tcgagctgac gtgtacttcc
1740
tatcgaagac gttagccgaa gcacctgtgt tcgccaccat accacttgtg ttcaccacca
1800
tagcatacta catgataggg ctgaaccctg aacctaagcg gttctttata gcgtccggtt
1860
tggctgccct gattactaac gttgctacgt cgtttggcta cctgatatcg tgtgccagca
1920
acagcgtgag catggcagcg tcagtgggac ctcccatcat catccccttc atgttgttcg
1980
gaggcttctt cctcaacact ggctccgtac caccatacct gggctggata tcgtacctgt
2040
cctggttcca ctacggcaac gaagcgctgc tggtcaacca gtggtctgga gtggaaacca
2100
tcgcctgcac ccgggagaac ttcacctgtc ccgcctctgg gcaggtcgtc ttggatactc
2160
ttagcttttc tgaggatgac ttcacaatgg acgtggtgaa catgatccta cttttcatcg
2220
gcttcagatt tttggcgtat ctcgctctct tgtaccgcgc tcgccgaggc aagtgagtct
2280
taggtacaaa atgctgcgag aatgggccat atgaaggaag aatgttgaat aaatagtgta
2340
attatttagg atgtaaggag tcaatggaga tttgataaat aaaacaattt ataccg
2396
<210> 128
<211> 1480
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая конструкцию кпРНК, направленно
воздействующую на ген ABC-переносчика white Helicoverpa
armigera
<400> 128
taatacgact cactataggg tatatgtcgg tccatccgca ccatagcctg gaatactaaa
60
tgctccttca cagtcagcgt gccaatgaac agatcctgct gctgaacata cgcagacagt
120
gctgataacg cctcaggggt agcaggctgg ccgttcagtg ctcgagtgcc actggacagc
180
accccgcttg gagtgcggaa ggtcagagtg ttgaggagtg tggtcttccc ggcaccggag
240
gatcccatga tggcgagcag ttcgcctggg taggcggctc catttacatt cctcaacaac
300
tgtttccttt gctggaacat gccactggcg gagggcttcc agaagttcca aatccttcgg
360
gacctagacc tggattcagt agcaaaggca ttgatgtctg cccaggtgta tgtgaccttc
420
tccacgcctc cgatggctcc atagttgttc cagcaaagga aacagttgtt gaggaatgta
480
aatggagccg cctacccagg cgaactgctc gccatcatgg gatcctccgg tgccgggaag
540
accacactcc tcaacactct gaccttccgc actccaagcg gggtgctgtc cagtggcact
600
cgagcactga acggccagcc tgctacccct gaggcgttat cagcactgtc tgcgtatgtt
660
cagcagcagg atctgttcat tggcacgctg actgtgaagg agcatttagt attccaggct
720
atggtgcgga tggaccgaca tatacccgta tgcgcagcgc atgaggagag ttcaagaggt
780
tattactgag ttggcgctaa caaaatgcca gaacacagtg ataggcatcc ctgggcggct
840
gaagggtatc tccggcgggg agatgaagag gctgtccttc gccagcgagg tgctcacgga
900
tccaccgctc atgttctgcg atgaacccac ctctggactc gattctttta tggcgcagaa
960
tgttatacag gtactgaaag gtctcgcaca aaaaggcaag acagtcgtat gcacgatcca
1020
ccagccgtct tcggagctgt acgcgatgat gaagtggtcc gctgggttgt agttaggagg
1080
acacgctgct cctaggtctt taaagaaatc attagcctga tcaggggagc cgaggaaggc
1140
gaccttcccg tctgccatga tgagcagctt atcgaacatc gcgtacagct ccgaagacgg
1200
ctggtggatc gtgcatacga ctgtcttgcc tttttgtgcg agacctttca gtacctgtat
1260
aacattctgc gccataaaag aatcgagtcc agaggtgggt tcatcgcaga acatgagcgg
1320
tggatccgtg agcacctcgc tggcgaagga cagcctcttc atctccccgc cggagatacc
1380
cttcagccgc ccagggatgc ctatcactgt gttctggcat tttgttagcg ccaactcagt
1440
aataacctct tgaactctcc tcatgcgctg cgcatacgta
1480
<210> 129
<211> 811
<212> ДНК
<213> Linepithema humile
<400> 129
agagagaacg atgaggacaa tgagatggaa aaaacaacaa cgtcccaacg tcccttcgac
60
gacgccattc caccagccct ataaaacccc gaggatcatc ggcgtcccaa cattactcgg
120
tcagagtctc gaggaacgcc gtgtccgaga tgatcatcac caggaaccgc atcaaccgcg
180
caactctaat ctgcgttctg gcgtcgtggc tttgcttggc gtctcgcgct tccgccgaat
240
acgaatcgcg ggagatgtcg aacggcggac cgggcgtcga cgcctcgtgc atcgagggca
300
agtgcatgaa gcgcaccgcc acgcaggatg ctaccgccag catgtggttc ggcccgcgtt
360
tgggaagacg gcgcagatcg gacgagaagc aggaagtgaa ttccgagata caggctctgg
420
cggaagcctt ggatagcggg cgtttggccc tatttgccat tccagctaac gacaagagac
480
aaccgactca atttacaccg cgactggggc gaggatcaga cgaggaccta tcctcctacg
540
gagacgcgat tgagaggaac gagatcgacg atcgtatatt acccgcgtta ttcgcgccgc
600
gtttaggacg acgaattcct tggtcaccgt cgccgagact tggacgccaa ttacgcagca
660
ttttgcgaaa aatgtaggcg ccgtcgaaag attattatca aaagttacaa atgaagagtg
720
atctcgtaga cctgcgcgtg aagatgaaat aacaactaaa attatagcac tattaagaca
780
taaagaaata aagtactgat gtttatttgt a
811
<210> 130
<211> 1360
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая конструкцию кпРНК, направленно
воздействующую на ген PBAN у аргентинских муравьев
<400> 130
taatacgact cactataggg aattcacttc ctgcttctcg tccgatctgc gccgtcttcc
60
caaacgcggg ccgaaccaca tgctggcggt agcatcctgc gtggcggtgc gcttcatgca
120
cttgccctcg atgcacgagg cgtcgacgcc cggtccgccg ttcgacatct cccgcgattc
180
gtattcggcg gaagcgcgag acgccaagca aagccacgac gccagaacgc agattagagt
240
tgcgcggttg atgcggttcc tggtgatgat catctcggac acggcgttcc tcgagactct
300
gaccgagtaa tgttgggacg ccgatgatcc tcggggtttt atagggctgg tggaatggcg
360
tcgtcgaagg gacgttggga cgttgttgtt ttttccatct cattgtcctc atcgttcacg
420
ccgtgtccga gatgatcatc accaggaacc gcatcaaccg cgcaactcta atctgcgttc
480
tggcgtcgtg gctttgcttg gcgtctcgcg cttccgccga atacgaatcg cgggagatgt
540
cgaacggcgg accgggcgtc gacgcctcgt gcatcgaggg caagtgcatg aagcgcaccg
600
ccacgcagga tgctaccgcc agcatgtggt tcggcccgcg tttgggaaga cggcgcagat
660
cggacgagaa gcaggaagtg aattcccgta atacaggctc tggcggaagc cttggatagc
720
gggcgtttgg ccctatttgc cattccagct aacgacaaga gacaaccgac tcaatttaca
780
ccgcgactgg ggcgaggatc agacgaggac ctatcctcct acggagacgc gattgagagg
840
aacgagatcg acgatcgtat attacccgcg ttattcgcgc cgcgtttagg acgacgaatt
900
ccttggtcac cgtcgccgag acttggacgc caattacgca gcattttgcg aaaaatgaaa
960
catcagtact ttatttcttt atgtcttaat agtgctataa ttttagttgt tatttcatct
1020
tcacgcgcag gtctacgaga tcactcttca tttgtaactt ttgataataa tctttcgacg
1080
gcgcctacat ttttcgcaaa atgctgcgta attggcgtcc aagtctcggc gacggtgacc
1140
aaggaattcg tcgtcctaaa cgcggcgcga ataacgcggg taatatacga tcgtcgatct
1200
cgttcctctc aatcgcgtct ccgtaggagg ataggtcctc gtctgatcct cgccccagtc
1260
gcggtgtaaa ttgagtcggt tgtctcttgt cgttagctgg aatggcaaat agggccaaac
1320
gcccgctatc caaggcttcc gccagagcct gtattacgta
1360
<210> 131
<211> 1480
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая конструкцию кпРНК, направленно
воздействующую на ген, кодирующий каталитическую субъединицу A
протонной АТФазы V-типа L. cuprina
<400> 131
taatacgact cactataggg aagttcttgt catagaaatc atccaaagca cgcatgtatt
60
tggagtagga aatcaaccaa ttgatggagg ggaaatgttt acgttgggcc aatttcttgt
120
ccaaacccca gaacacttgt acgataccca aagtggcaga agtaacggga tcagagaaat
180
caccaccagg aggagataca gcaccgacaa tggaaacgga accttcacgt tcagggttac
240
ccaaacactt gacacgacca gcacgttcgt agaaggaggc caaacgggca cccaagtagg
300
ctgggtaacc ggaatcggca ggcatttcag ccaaacgacc agaaatttca cgaagagctt
360
cggcccaacg ggaggtagaa tcagccatca tagatacgtt gtaacccata tcacggaagt
420
attcagacaa ggtaataccg gtataaacga ttccggttac ccagcctact tgggtgcccg
480
tttggcctcc ttctacgaac gtgctggtcg tgtcaagtgt ttgggtaacc ctgaacgtga
540
aggttccgtt tccattgtcg gtgctgtatc tcctcctggt ggtgatttct ctgatcccgt
600
tacttctgcc actttgggta tcgtacaagt gttctggggt ttggacaaga aattggccca
660
acgtaaacat ttcccctcca tcaattggtt gatttcctac tccaaataca tgcgtgcttt
720
ggatgatttc tatgacaaga acttcccgta attcgtacca ttgcgtacca aggtcaagga
780
aatcttgcaa gaagaagaag atttgtccga aattgtacaa ttggtcggta aggcttcatt
840
ggccgaaact gacaagatca ccttggaagt cgccaaattg cttaaggacg atttcttgca
900
acagaactcc tactcatcat acgacagatt ctgccccttc tacaagagtg tgggtatgtt
960
gaagaacatc attgccttct acgacttggc tcgtcactcc gtcgaatcca ccgctcaatc
1020
tgaaaacaaa atcacctgga atgtcattct gaaagcctgt tgtaaatctt cgtgtaattg
1080
ttcaaagtca gccttgatct tggcttcacc gtccttaacg ggatccttga atttcatgga
1140
agacaattgg tacataatgt tacccatagc ttcacggatg acattccagg tgattttgtt
1200
ttcagattga gcggtggatt cgacggagtg acgagccaag tcgtagaagg caatgatgtt
1260
cttcaacata cccacactct tgtagaaggg gcagaatctg tcgtatgatg agtaggagtt
1320
ctgttgcaag aaatcgtcct taagcaattt ggcgacttcc aaggtgatct tgtcagtttc
1380
ggccaatgaa gccttaccga ccaattgtac aatttcggac aaatcttctt cttcttgcaa
1440
gatttccttg accttggtac gcaatggtac gaattacgta
1480
<210> 132
<211> 1480
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая конструкцию кпРНК, направленно
воздействующую на ген, кодирующий РНКазу 1/2 L. cuprina
<400> 132
taatacgact cactataggg aataatttgt ggtagacata gcgggttact tcctcatgtt
60
cgttaaagca gacctggtat tgtctcatga aacgggaaga tgacaattca aaaccaacat
120
tgacgagagt agtgccacca ttgcagctgc tgcccgactt cttagctaca aaagcaggcc
180
aactggtgca aacaagactg ggcagggagt gctgaacacc attgacttta aaagtggtgc
240
cactaacaca ggtagcgata tgggtcttgc ctgataaagg atgagcaaag ccactggtac
300
aatggatctc aatattcttg ccagcagcca catcaattct tccagtatca gaaaaagggt
360
agagttcagt agtgccaggt ttgatataca agggttgttt agccttaaga ccaccgcgaa
420
tgggtatgga acaaccacca ctgcgtggaa tattgagatc cattgtacca gtggctttgc
480
tcatccttta tcaggcaaga cccatatcgc tacctgtgtt agtggcacca cttttaaagt
540
caatggtgtt cagcactccc tgcccagtct tgtttgcacc agttggcctg cttttgtagc
600
taagaagtcg ggcagcagct gcaatggtgg cactactctc gtcaatgttg gttttgaatt
660
gtcatcttcc cgtttcatga gacaatacca ggtctgcttt aacgaacatg aggaagtaac
720
ccgctatgtc taccacaaat tattcccgta cccaacagcg tgccactttc ctattcatta
780
atgcagctcc ccagtggcaa gttttcaatg ccggtaattg ggctcgtgta gaggatggtg
840
tacgcgcctg ggtgtccaaa aataaaatca atgttcgatg ctataccggt gtttatggtg
900
tcaccactct acccaacaaa gagggacgtg agactcctct atatttgtct cgtgatgcca
960
ataataatgg tttgattcct gttcccaaat tatacttccg tgtggttata caacctgcca
1020
ccaataaggg tattgttttc gttggtgtca caggcataag aataaccagc agtgatatca
1080
gttttcttcc aactaatata gttaacctta tcactgacat ctttgcaaat aatatagtcc
1140
tttttgattt gttccaaagt caaatgggga ttgttgacac caacgaaaac aataccctta
1200
ttggtggcag gttgtataac cacacggaag tataatttgg gaacaggaat caaaccatta
1260
ttattggcat cacgagacaa atatagagga gtctcacgtc cctctttgtt gggtagagtg
1320
gtgacaccat aaacaccggt atagcatcga acattgattt tatttttgga cacccaggcg
1380
cgtacaccat cctctacacg agcccaatta ccggcattga aaacttgcca ctggggagct
1440
gcattaatga ataggaaagt ggcacgctgt tgggtacgta
1480
<210> 133
<211> 1480
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая конструкцию кпРНК, направленно
воздействующую на ген, кодирующий хитинсинтазу L. cuprina
<400> 133
taatacgact cactataggg tggcttgatt tttatattaa caccatacac ctcaaatgca
60
gctttttcaa tgctggtaat caatattttt acatattcat tgagaggagg atttttcgga
120
ttctccacat atttttcatc cagtataaag gcatcatcaa agaaaatatt agctaaaatg
180
taaaaaaaag aaaacttaat tgatagatac tcttcattcc aatccacaat tcgtctattt
240
aatgttatac attgttcaac caaaaaacca caataccacg gacaaatgaa cagtttttcg
300
gtgggtaaat ttttatcatt ttttggacgc catatatgat ttgttatcca caattgtgac
360
agccaccaca acaaccatat ccaaagataa tctttagcga ccacattaaa aaaactccat
420
gtatcgtgac cgaagcccag tgacgttgat aaaaatttac ccaccgaaaa actgttcatt
480
tgtccgtggt attgtggttt tttggttgaa caatgtataa cattaaatag acgaattgtg
540
gattggaatg aagagtatct atcaattaag ttttcttttt tttacatttt agctaatatt
600
ttctttgatg atgcctttat actggatgaa aaatatgtgg agaatccgaa aaatcctcct
660
ctcaatgaat atgtaaaaat attgattacc agcattgaaa aagctgcatt tgaggtgtat
720
ggtgttaata taaaaatcaa gccacccgta aaaattgaaa caccttatgg cggtcgtttg
780
gtgtggacac tgcctggtcg ctcaaagatg attgcccatt taaaaaacaa agataaaata
840
cgacataaga aacgctggtc acaggttatg tacatgtact atttgttggg ttttcgtata
900
atggaattgg aatcagtatc ggccaagcgt aaggcagtga tagcagaaaa tacatttttg
960
ctggctcttg atggtgatat tgactttcaa ccgcaggcag tgcaactgtt aatagaccgt
1020
atgaaggcca tagatgaatt aggtgctagc caggactaca taaaacacaa ccaataacat
1080
gctctgttgc tttttgcaac caatgaccta tagcgtattc gaagatttga taccaaacca
1140
tagggcctct accaactgga tgaatacgac cacaggcagc acctaattca tctatggcct
1200
tcatacggtc tattaacagt tgcactgcct gcggttgaaa gtcaatatca ccatcaagag
1260
ccagcaaaaa tgtattttct gctatcactg ccttacgctt ggccgatact gattccaatt
1320
ccattatacg aaaacccaac aaatagtaca tgtacataac ctgtgaccag cgtttcttat
1380
gtcgtatttt atctttgttt tttaaatggg caatcatctt tgagcgacca ggcagtgtcc
1440
acaccaaacg accgccataa ggtgtttcaa tttttacgta
1480
<210> 134
<211> 1481
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая конструкцию кпРНК, направленно
воздействующую на ген, кодирующий рецептор экдизона (EcR) L.
cuprina
<400> 134
taatacgact cactataggg tgaaagatca tcacgacctg atgatatgga attagctgat
60
tcagatctgg gtgtatgatg catcatacta ctgttattca tatgatgatg gtgatgatga
120
tttaatccat tactgatgat actgtgaatg ccaatattgg catgaataac ttggccattt
180
tgtgaggcct gtaacgagtt taactgttgg gcattgctaa cgatattggg accattaata
240
ttgaccgata agccaccacc accgccgcca atacccatgt ggccatttgt gtggtgggaa
300
ctgctattac tgtgattact gttgctgttg tggtgtaaat gattgtggct gtgattgtga
360
ttattcactt gactgccacc accaccaccc agaccattga gtgaagtcat accgggtaca
420
ccaccaccac ctccacctcc tccaacaaat cacagtaata gcagttccca ccacacaaat
480
ggccacatgg gtattggcgg cggtggtggt ggcttatcgg tcaatattaa tggtcccaat
540
atcgttagca atgcccaaca gttaaactcg ttacaggcct cacaaaatgg ccaagttatt
600
catgccaata ttggcattca cagtatcatc agtaatggat taaatcatca tcaccatcat
660
catatgaata acagtagtat gatgcatcat acacccagat ctgaatcagc taattccata
720
tcatcaggtc gtgatgatct ttcacccgta tccaccaaat caccccctta gtggttcgaa
780
acacttgtgt tccatttgtg gagaccgcgc cagtggaaaa cattatgggg tctacagttg
840
tgagggttgt aaagggttct tcaaacgtac cgtacgcaag gacttgacat atgcttgtcg
900
tgaggacaga aattgcatta tagataaacg acaaagaaat cgttgccagt attgtcgtta
960
tcaaaagtgt ttagcttgtg gcatgaaacg cgaagcggtc caagaggaac gacaacgtgg
1020
tactcgtgct gctaacgcta gagctgcctt ttgctcggct tcaatgatgc gttctatagt
1080
gagatcacgt aatgaactgc tgggtttaaa gtcttctccg ccagcaccaa ccacattgct
1140
taccccacca ccacctcctc caccaccgcc agcaccagca gctctagcgt tagcagcacg
1200
agtaccacgt tgtcgttcct cttggaccgc ttcgcgtttc atgccacaag ctaaacactt
1260
ttgataacga caatactggc aacgatttct ttgtcgttta tctataatgc aatttctgtc
1320
ctcacgacaa gcatatgtca agtccttgcg tacggtacgt ttgaagaacc ctttacaacc
1380
ctcacaactg tagaccccat aatgttttcc actggcgcgg tctccacaaa tggaacacaa
1440
gtgtttcgaa ccactaaggg ggtgatttgg tggatacgta g
1481
<210> 135
<211> 1481
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая конструкцию кпРНК, направленно
воздействующую на ген, кодирующий гамма-тубулин 1/1-подобный
белок
L. cuprina
<400> 135
taatacgact cactataggg aaaacgctgt aggtttgtat aagtttctta ggaaaacgat
60
cagacaaacg ttccataata taagagccca tgccggaacc agtaccaccg gctatagaat
120
ggcatagaac aaatccctcc aaggaatcac tgccatctgc ctcacgatca ataatgtcaa
180
aaatttcctc ttgtaatttt tcaccttgac tatagccgga agcccaattg ttgccggcac
240
caccaccatg tttagacaag taaacatttt cgggattata taacttggca tagggtgaac
300
tcataatggt gtgtataact cgcggctcca aatccaaaag tacggcacgt ggtatatagt
360
gatcatcgtc agcctgataa aagaatacat ccttgcgatc tactccatct gtagcaaaat
420
cctctaacac tccactaggt gaaatgctat acacaccatt atgagttcac cctatgccaa
480
gttatataat cccgaaaatg tttacttgtc taaacatggt ggtggtgccg gcaacaattg
540
ggcttccggc tatagtcaag gtgaaaaatt acaagaggaa atttttgaca ttattgatcg
600
tgaggcagat ggcagtgatt ccttggaggg atttgttcta tgccattcta tagccggtgg
660
tactggttcc ggcatgggct cttatattat ggaacgtttg tctgatcgtt ttcctaagaa
720
acttatacaa acctacagcg ttttcccgta ccacaaccct acgttatcct tcatatatga
780
ataataattt gataggattg acggcacctt tgatacctac cccccaatta cattttctaa
840
tgaccggtta tactcctcta actacagata gtgatcccaa tttgaatata cgcaaaacta
900
cggtactaga tgttatgaga cgtttattgc aacccaaaaa tatgatggtt tcatcgggtc
960
cggataaagc aaatattcat tgttatattt ccatattaaa tattatacag ggtgaagtag
1020
atcccactca agtccacaaa tctctactga ttggccatca ttaggcccga aactttatga
1080
ttactttgta tatatggaga acttctggac aaggctactt gtatactggc cggaccccag
1140
ggtatgaatt gagctaattt gcgttcacgt atacgttgta gagatttgtg gacttgagtg
1200
ggatctactt caccctgtat aatatttaat atggaaatat aacaatgaat atttgcttta
1260
tccggacccg atgaaaccat catatttttg ggttgcaata aacgtctcat aacatctagt
1320
accgtagttt tgcgtatatt caaattggga tcactatctg tagttagagg agtataaccg
1380
gtcattagaa aatgtaattg gggggtaggt atcaaaggtg ccgtcaatcc tatcaaatta
1440
ttattcatat atgaaggata acgtagggtt gtggtacgta g
1481
<210> 136
<211> 1605
<212> ДНК
<213> Triticum aestivum
<400> 136
atggcaaagg acgacgggta ccccccggcg cggacgctgc cggagacgcc gtcctgggcg
60
gtggcgctgg tcttcgccgt catgatcatc gtctccgtcc tcctggagca cgcgctccac
120
aagctcggcc attggttcca caagcggcac aagaacgcgc tggcggaggc gctggagaag
180
atgaaggcgg agctgatgct ggtgggattc atctcgctgc tgctcgccgt cacgcaggac
240
ccaatctccg ggatatgcat ctcccagaag gccgccagca tcatgcgccc ctgcaaggtg
300
gaacccggtt ccgtcaagag caagtacaag gactactact gcgccaaaga gggcaaggtg
360
gcgctcatgt ccacgggcag cctgcaccag ctccacatat tcatcttcgt gctagccgtc
420
ttccatgtca cctacagcgt catcatcatg gctctaagcc gtctcaagat gagaacatgg
480
aagaaatggg agacagagac cgcctccttg gaataccagt tcgcaaatga tcctgcgcgg
540
ttccgcttca cgcaccagac gtcgttcgtg aagcggcacc tgggcctgtc cagcaccccc
600
ggcgtcagat gggtggtggc cttcttcagg cagttcttca ggtcggtcac caaggtggac
660
tacctcatct tgagggcagg cttcatcaac gcgcacttgt cgcagaacag caagttcgac
720
ttccacaagt acatcaagag gtccatggag gacgacttca aagtcgtcgt tggcatcagc
780
ctcccgctgt gggctgtggc gatcctcacc ctcttccttg atatcgacgg gatcggcaca
840
ctcacctggg tttctttcat ccctctcatc atcctcttgt gtgttggaac caagctagag
900
atgatcatca tggagatggc cctggagatc caggaccggt cgagcgtcat caagggggca
960
cccgtggtcg agcccagcaa caagttcttc tggttccacc gccccgactg ggtcctcttc
1020
ttcatacacc tgacgctgtt ccagaacgcg tttcagatgg cacatttcgt gtggacagtg
1080
gccacgcccg gcttgaagga ctgcttccat atgaacatcg ggctgagcat catgaaggtc
1140
gtgctggggc tggctctcca gttcctgtgc agctacatca ccttccccct ctacgcgcta
1200
gtcacacaga tgggatcaaa catgaagagg tccatctttg acgagcagac agccaaggcg
1260
ctgaccaact ggcggaacac ggccaaggag aagaagaagg tccgagacac ggacatgctg
1320
atggcgcaga tgatcggcga cgcaacaccc agccgaggca cgtccccgat gcctagccgg
1380
ggctcatcgc cggtgcacct gcttcagaag ggcatgggac ggtctgacga tccccagagc
1440
gcaccgacct cgccaaggac catggaggag gctagggaca tgtacccggt tgtggtggcg
1500
catcctgtac acagactaaa tcctgctgac aggcggaggt cggtctcttc atcagccctc
1560
gatgccgaca tccccagcgc agatttttcc ttcagccagg gatga
1605
<210> 137
<211> 1277
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая конструкцию кпРНК, направленно
воздействующую на Mlo из Triticum aestivum
<400> 137
taatacgact cactataggg tgcccccttg atgacgctcg accggtcctg gatctccagg
60
gccatctcca tgatgatcat ctctagcttg gttccaacac acaagaggat gatgagaggg
120
atgaaagaaa cccaggtgag tgtgccgatc ccgtcgatat caaggaagag ggtgaggatc
180
gccacagccc acagcgggag gctgatgcca acgacgactt tgaagtcgtc ctccatggac
240
ctcttgatgt acttgtggaa gtcgaacttg ctgttatgcg acaaatgcgc gttgatgaag
300
cctgccctca aggtgaggta gtccaccttg gtgaccgacc tgaagaactg cctgaagaag
360
gccaccaccc atctgacgcc gggggtgctg gagaggggtt cgacttccac aagtacatca
420
agaggtccat ggaggacgac ttcaaagtcg tcgttggcat cagcctcccg ctgtgggctg
480
tggcgatcct caccctcttc cttgatatcg acgggatcgg cacactcacc tgggtttctt
540
tcatccctct catcatcctc ttgtgtgttg gaaccaagct agagatgatc atcatggaga
600
tggccctgga gatccaggac cggtcgagcg tcatcaaggg ggcacccgac gtcgagccca
660
gcaacaagtt cttctggttc caccgccccg actgggtcct cttcttcata cacctgacgc
720
tgttccagaa gtcacacaga tgggatcaaa catgaagagg tccatcttcg acgagcagac
780
agccaaggcg ctgaccaact ggcggaacac ggccaaggag aagaagaagg tccgagacac
840
ggacatgctg atggcgcaga tgatcggcga cgcgacgccc agccgaggca cgtcccccac
900
cacaaccggg tacatgtccc tagcctcctc catggtcctt ggcgaggtcg gtgcgctctg
960
gggatcgtca gaccgtccca tgcccttctg aagcaggtgc accggcgatg agccccggct
1020
aggcatcggg gacgtgcctc ggctgggcgt cgcgtcgccg atcatctgcg ccatcagcat
1080
gtccgtgtct cggaccttct tcttctcctt ggccgtgttc cgccagttgg tcagcgcctt
1140
ggctgtctgc tcgtcgaaga tggacctctt catgtttgat cccatctgtg tgacttctgg
1200
aacagcgtca ggtgtatgaa gaagaggacc cagtcggggc ggtggaacca gaagaacttg
1260
ttgctgggct cgacgtc
1277
<210> 138
<211> 1527
<212> ДНК
<213> Vitis pseudoreticulata
<400> 138
atggctggcg acgaggagac gacgacgacg gcagcaacac ttgaaacaac gtccacttgg
60
gctgttgcct ctgtttgctt tattttgatt gcactctcca tacttattga gcatgccctc
120
catctcttag ccaagtactt caacaagaag cggaggaggt ctctcattca tgctcttaac
180
aacgtcaaat cggagttgat gctcttgggg ttcgtctctt tgttgctgac tgtgtgccaa
240
aagtatattg cgaagatttg tatcccaagg agcgtaggtg aaacttttct tccctgcaag
300
accttgacag aaagtgattc agaagaagaa accaaatgcg aagagcaggg aaagatgtct
360
ttgctgtcta gacaaggcgt ggaggaacta caatacttaa ttttcgtgct ggccttcttc
420
cattccctct actgcgtcct cacattcggt cttgggatgg ccaagatgaa gaaatgggag
480
tcctgggagg cagaaacaag aacactggaa tatcagttta caaatgatcc acggaggttc
540
aggctcatcc atcagacatc atttggaaag caacatctga gatattggag tgagcatcag
600
atacttcgtt ggccggcttg ttttattcgg cagttctatc catccgtctc caaagtggat
660
tacttgactc ttagacatgg gttcattatg gcccattttg cagaaggaag caactatgac
720
ttccaaaagt atataaaaag agctttggaa aaagactttg gagtggtggt gggaggaagt
780
ttctgggttt ggagtttctc catgcttttt gtgttcttca atgctcaagt attttacaac
840
tatttatggc taccctttat tccattggtg atgctgttgt tggttggaac aaagctacag
900
ggcattataa ctaagatgtg cttagatagc catgataaag ctctcgttgt tagaggaact
960
ttgcttgtca ggcccagtga tcacttcttc tggtttggaa aaccggaatt gctcctacat
1020
cttatgcact ttatattgtt tcagaactct tttcaactgg cgttctttac atggacttgg
1080
tacaaatttg gattcagatc atgcttccat gatacaactg aggatatcgt cataaggctt
1140
gtcatgggtg tgttagtaca actcctttgt ggctacgtga cactgcctct gtatgccctg
1200
gtcacgcaga tggggacatc aatgaggaca attgtcttta ctgagggagt cgttgaaggt
1260
ctgaacagat ggagaaggaa agccaagaaa aacatagcac gcaggaacaa ccactcagct
1320
cgtccctccc tggatgcttc actcgacaat tcaccttctt ttaacactct ggatacttct
1380
ttctctgtag acctcgatca gccatcatca gatgctggtt atttgactgt tgaaatatca
1440
gatgaagaga cggtcgctac taaacggcca gaaccgcgtc agaagttggg atcttttgag
1500
ggtttcgact cgtgcaaaac atcataa
1527
<210> 139
<211> 1480
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Химерная ДНК, кодирующая конструкцию кпРНК, направленно
воздействующую на ген MLO Vitis
<400> 139
taatacgact cactataggg tagccataaa tagttgtaaa atacttgagc attgaagaac
60
acaaaaagca tggagaaact ccaaacccag aaacttcctc ccaccaccac tccaaagtct
120
ttttccaaag ctctttttat atacttttgg aagtcatagt tgcttccttc tgcaaaatgg
180
gccataatga acccatgtct aagagtcaag taatccactt tggagacgga tggatagaac
240
tgctgaataa aacaaaccgg ccaacgaagt atccgatgct cactccaata tctcagatgt
300
tgctttccaa atgatgtctg atggatgagc gtgaacctcc gtggatcatt tgtaaactga
360
tattccagtg ttcttgtttc tgcctcccag gactcccatt tcttcatctt ggccatccca
420
agaccgaatg tgaggacgca gtagagcaca tcatttggaa agcaacatct gagatattgg
480
agtgagcatc ggatacttcg ttggccggtt tgttttattc agcagttcta tccatccgtc
540
tccaaagtgg attacttgac tcttagacat gggttcatta tggcccattt tgcagaagga
600
agcaactatg acttccaaaa gtatataaaa agagctttgg aaaaagactt tggagtggtg
660
gtgggaggaa gtttctgggt ttggagtttc tccatgcttt ttgtgttctt caatgctcaa
720
gtattttaca actatttatg gctacccgta attccattgg tgatgctgtt gttggttgga
780
acaaagctac agggcattat aactaagatg tgcctagata gccatgataa agctctcgtt
840
gttagaggaa ctttgcttgt caggcccagt gatcacttct tctggtttgg aaaaccggaa
900
ttgctcctac atcttatgca ctttatattg tttcagaact cttttcaact ggcgttcttt
960
acatggactt ggtacaaatt tggattcaga tcatgcttcc atgatacaac tgaggatatc
1020
gtcataaggc ttgtcatggg tgtgttatgg ctttccttct ccatctgttc agaccttcaa
1080
cgactccctc agtaaagaca attgtcctca ttgatgtccc catctgcgtg accagggcat
1140
acagaggcag tgtcacgtag ccacaaagga gttgtactaa cacacccatg acaagcctta
1200
tgacgatatc ctcagttgta tcatggaagc atgatctgaa tccaaatttg taccaagtcc
1260
atgtaaagaa cgccagttga aaagagttct gaaacaatat aaagtgcata agatgtagga
1320
gcaattccgg ttttccaaac cagaagaagt gatcactggg cctgacaagc aaagttcctc
1380
taacaacgag agctttatca tggctatcta ggcacatctt agttataatg ccctgtagct
1440
ttgttccaac caacaacagc atcaccaatg gaattacgta
1480
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| МОЛЕКУЛЫ РНК, ВКЛЮЧАЮЩИЕ НЕКАНОНИЧЕСКИЕ ПАРЫ ОСНОВАНИЙ | 2019 |
|
RU2812710C2 |
| МОЛЕКУЛЫ НУКЛЕИНОВОЙ КИСЛОТЫ, КОТОРЫЕ ВОЗДЕЙСТВУЮТ НА СУБЪЕДИНИЦУ С ВАКУОЛЯРНОЙ АТФАЗЫ И ПРИДАЮТ УСТОЙЧИВОСТЬ К ЖЕСТКОКРЫЛЫМ НАСЕКОМЫМ-ВРЕДИТЕЛЯМ | 2011 |
|
RU2644669C2 |
| МОЛЕКУЛЫ НУКЛЕИНОВЫХ КИСЛОТ, КОТОРЫЕ ПРИДАЮТ УСТОЙЧИВОСТЬ К НАСЕКОМЫМ-ВРЕДИТЕЛЯМ ОТРЯДА ЖЕСТКОКРЫЛЫХ | 2011 |
|
RU2639549C2 |
| СПОСОБЫ ГЕНЕТИЧЕСКОГО КОНТРОЛЯ ПОРАЖЕНИЯ РАСТЕНИЙ НАСЕКОМЫМИ И ПРИМЕНЯЕМЫЕ ДЛЯ ЭТОГО КОМПОЗИЦИИ | 2006 |
|
RU2478710C2 |
| ЛЕЧЕНИЕ НАРУШЕНИЙ ЦНС | 2007 |
|
RU2426544C2 |
| УМЕНЬШЕНИЕ ЭКСПРЕССИИ ГЕНОВ У НАСЕКОМЫХ-ВРЕДИТЕЛЕЙ | 2011 |
|
RU2662995C2 |
| КОМПОЗИЦИИ И СПОСОБЫ БОРЬБЫ С LEPTINOTARSA | 2014 |
|
RU2703498C2 |
| ОЛИГОРИБОНУКЛЕОТИДЫ И СПОСОБЫ ИХ ПРИМЕНЕНИЯ ДЛЯ ЛЕЧЕНИЯ ОСТРОЙ ПОЧЕЧНОЙ НЕДОСТАТОЧНОСТИ И ДРУГИХ ЗАБОЛЕВАНИЙ | 2005 |
|
RU2434942C2 |
| КОМПОЗИЦИИ И СПОСОБЫ ДЛЯ МОДИФИКАЦИИ ЗАДАННОЙ ПОСЛЕДОВАТЕЛЬНОСТИ НУКЛЕИНОВОЙ КИСЛОТЫ-МИШЕНИ | 2012 |
|
RU2663354C2 |
| ОЛИГОРИБОНУКЛЕОТИДЫ И СПОСОБЫ ИХ ПРИМЕНЕНИЯ ДЛЯ ЛЕЧЕНИЯ ОСТРОЙ ПОЧЕЧНОЙ НЕДОСТАТОЧНОСТИ | 2005 |
|
RU2584609C2 |
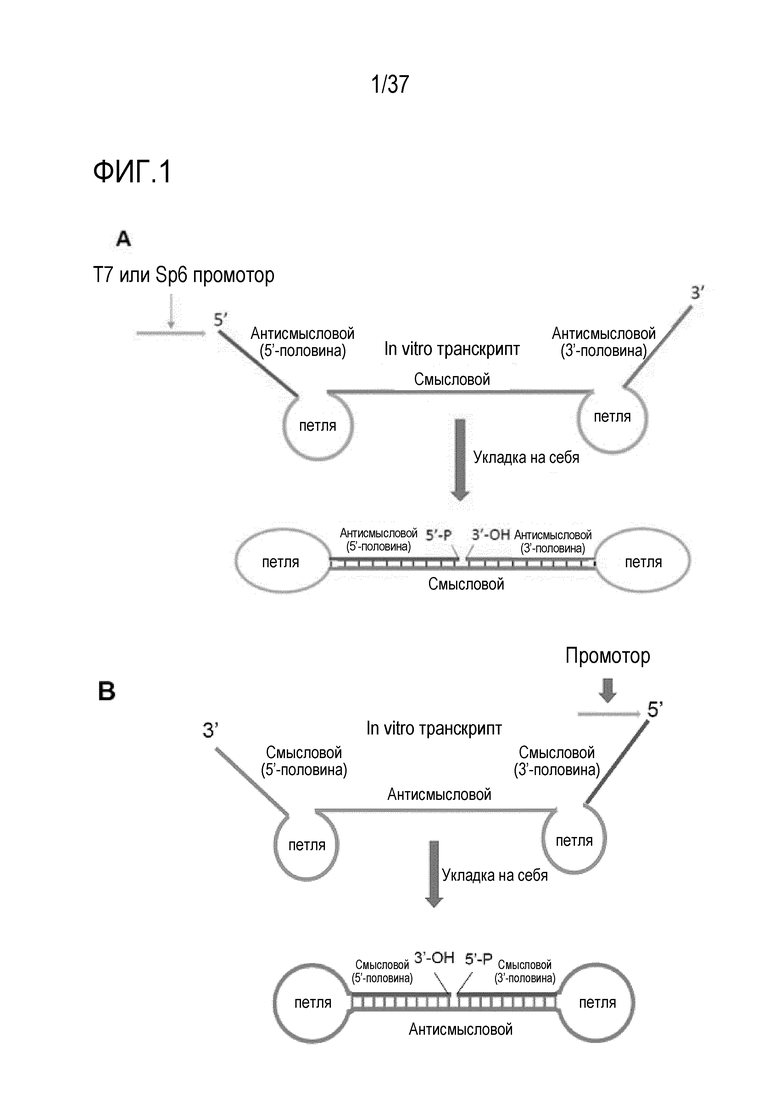

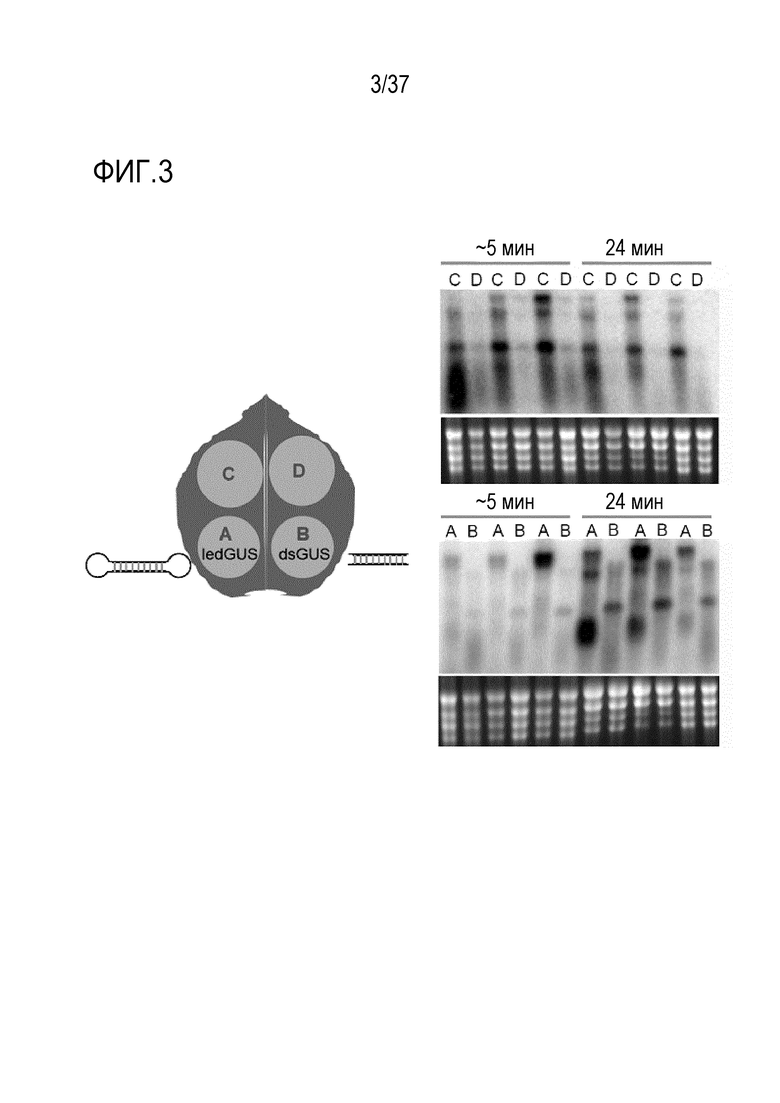






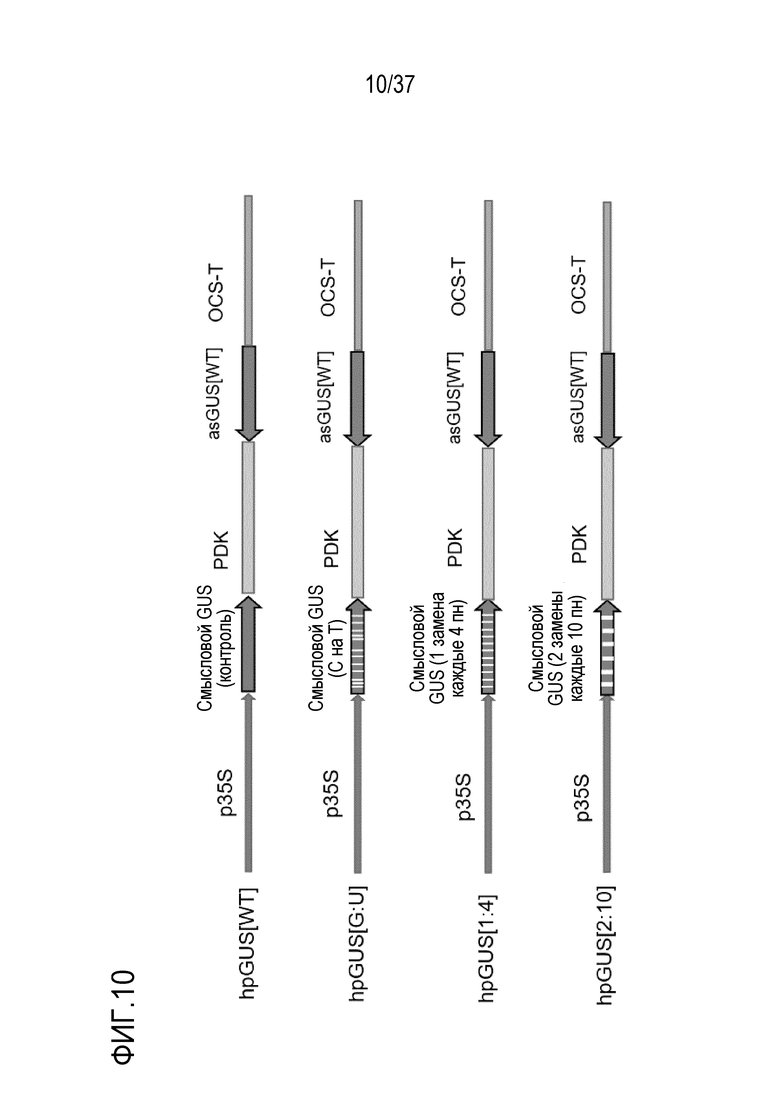

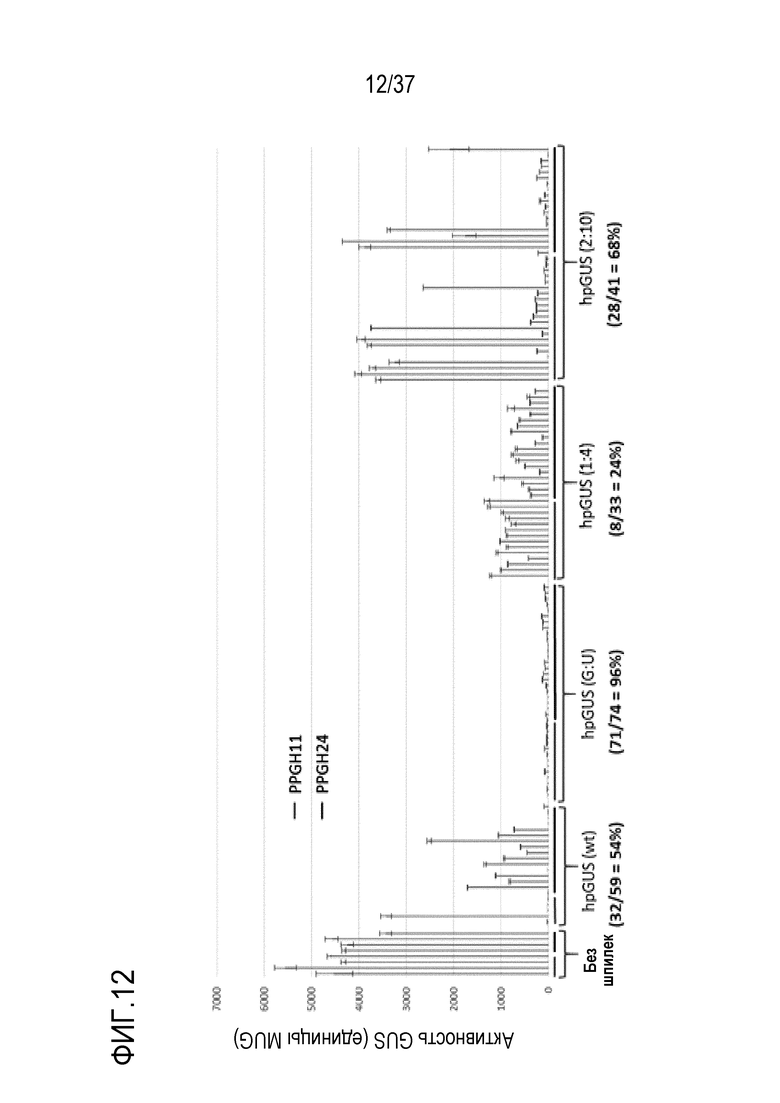

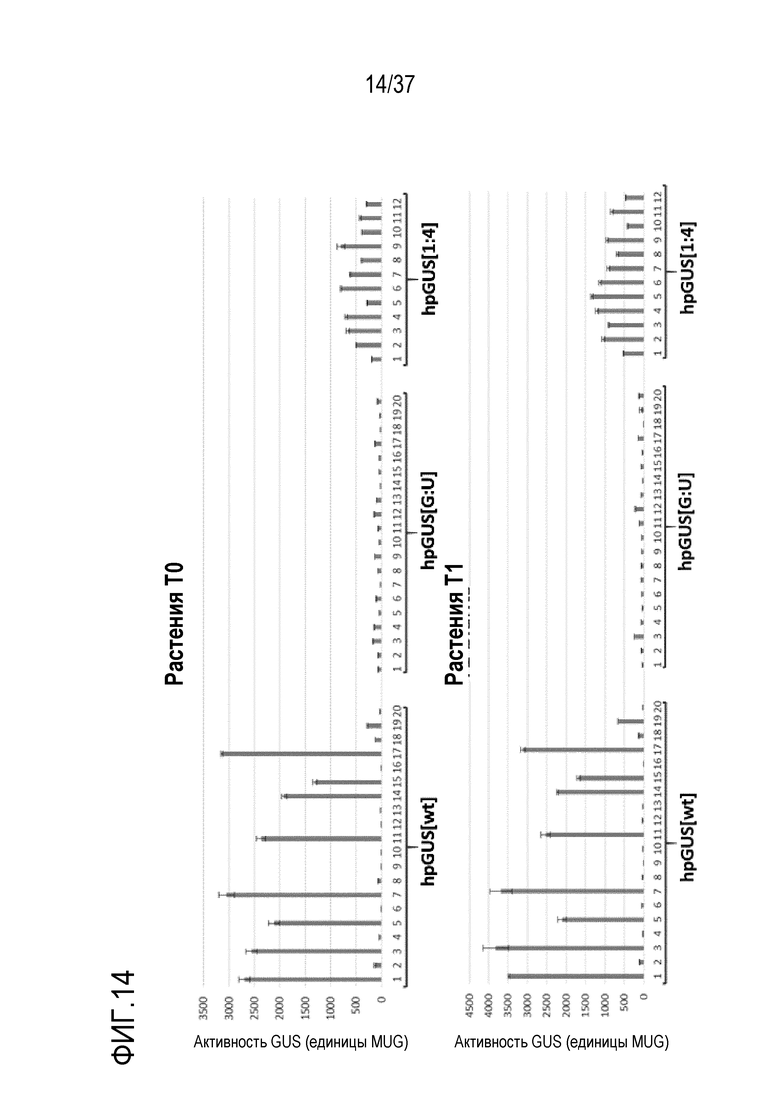












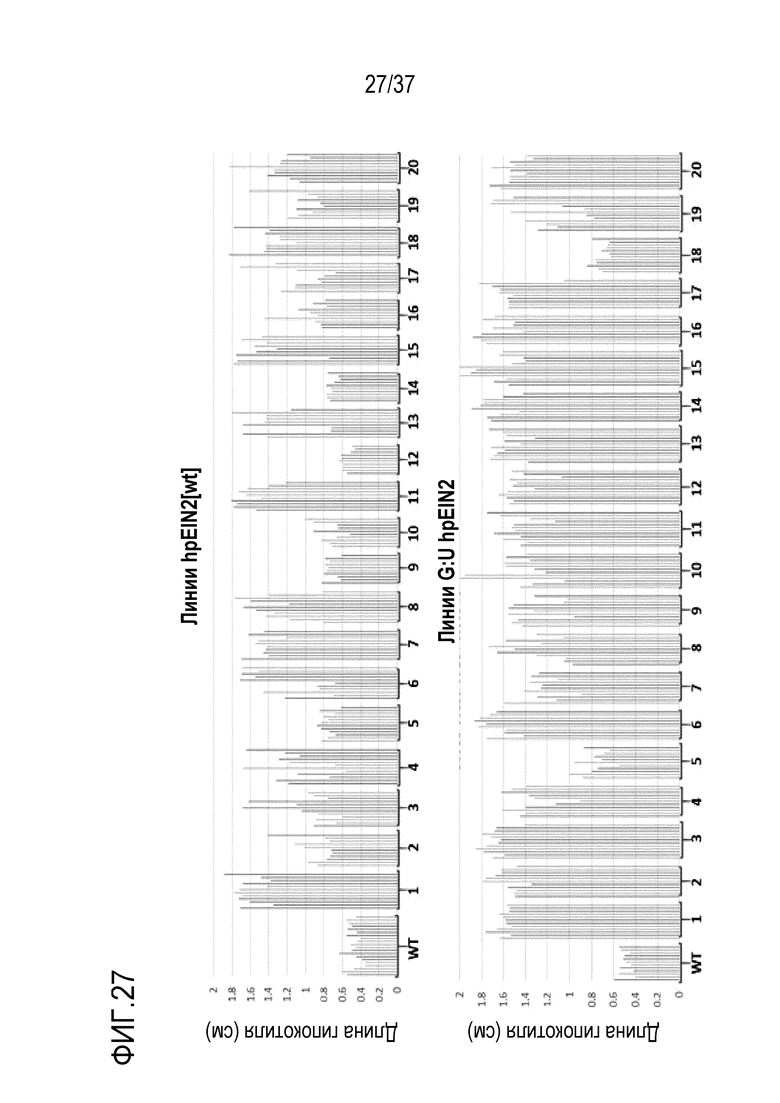





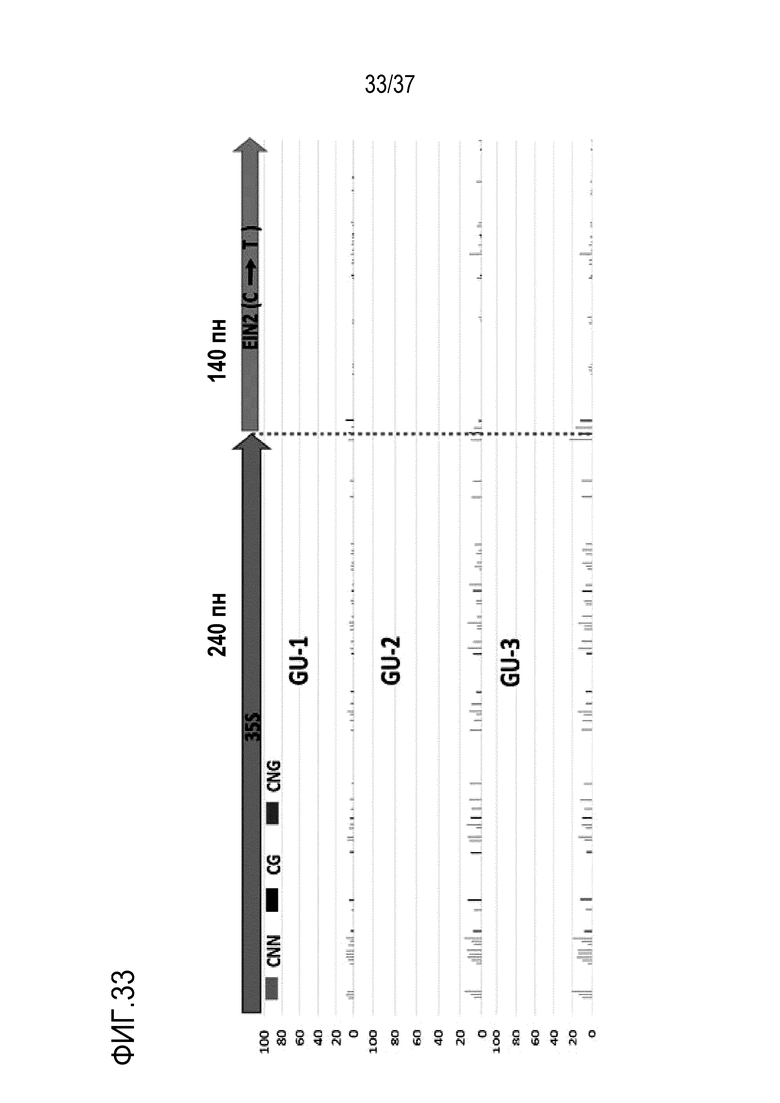
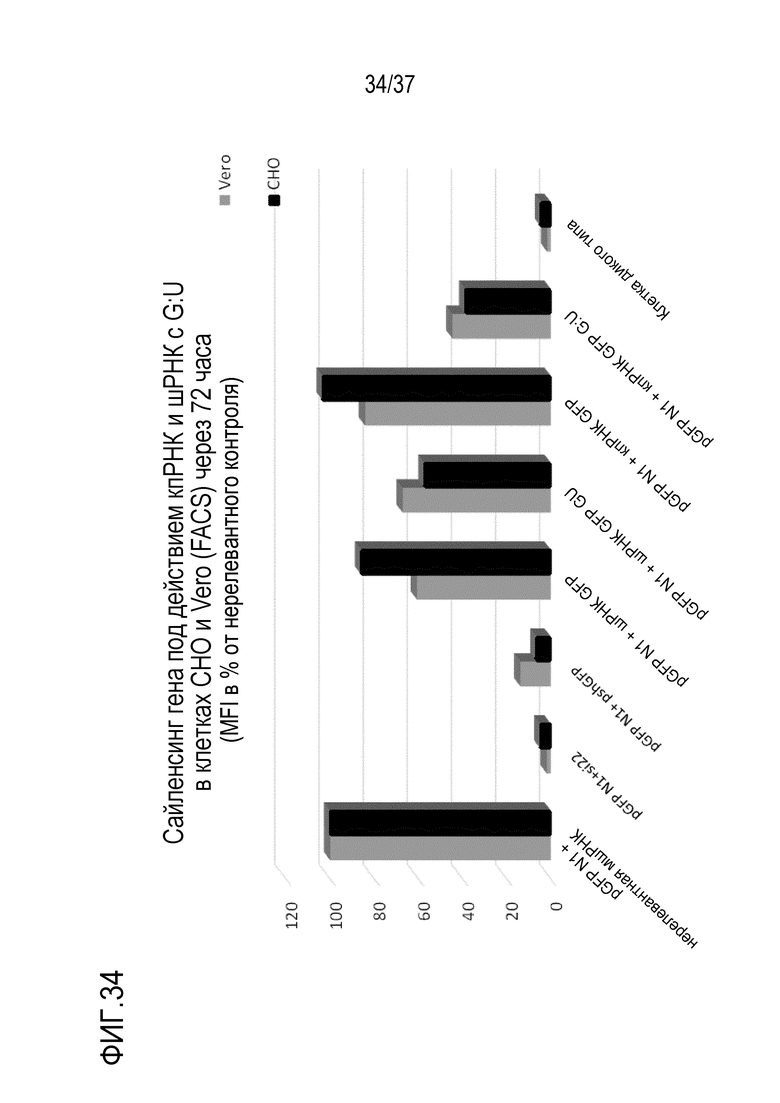
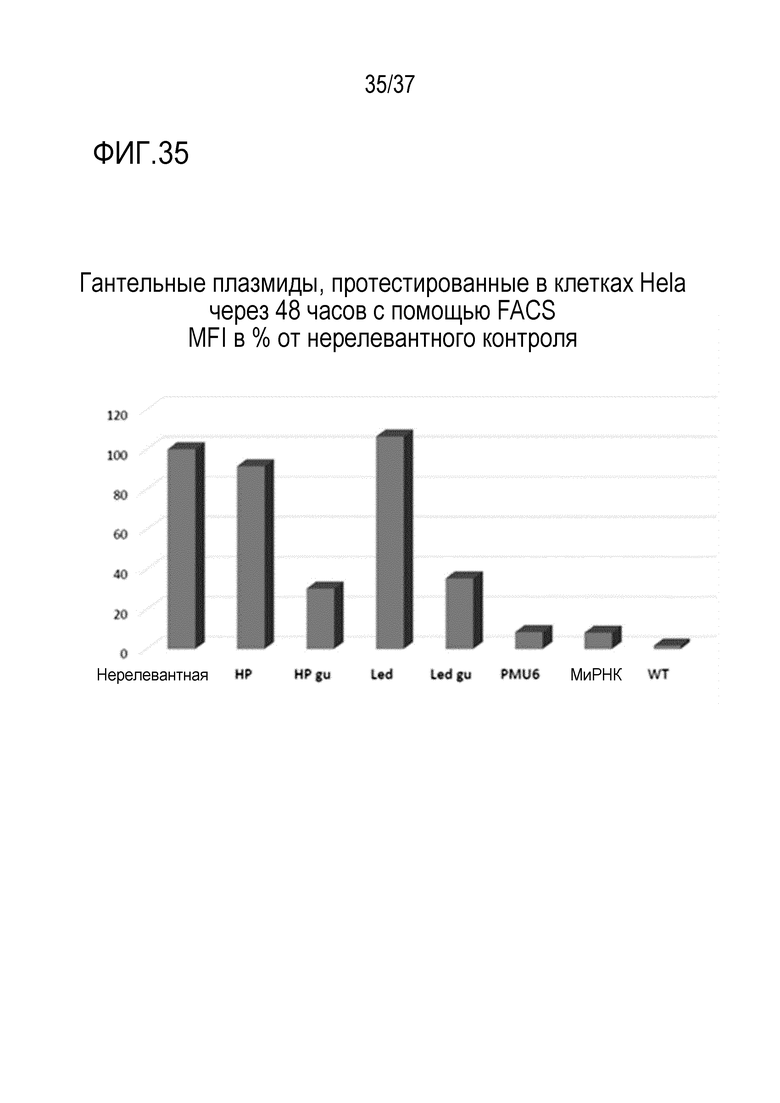


Изобретение относится к биотехнологии и представляет собой молекулу РНК для даунрегуляции уровня и/или активности молекулы РНК-мишени, где молекула РНК включает первый РНК-компонент, второй РНК-компонент, который ковалентно связан с первым РНК-компонентом, и, необязательно, одно или более, или все из: (i) соединительной рибонуклеотидной последовательности, которая ковалентно связывает первый и второй РНК-компоненты, (ii) 5'-лидерной последовательности и (iii) 3'-трейлерной последовательности, где первый РНК-компонент состоит, в порядке от 5' к 3', из первого 5'-рибонуклеотида, первой последовательности РНК и первого 3'-рибонуклеотида, где первые 5' и 3'-рибонуклеотиды спариваются друг с другом в первом РНК-компоненте, где первая последовательность РНК включает первую смысловую рибонуклеотидную последовательность по меньшей мере из 20 последовательных рибонуклеотидов, первую последовательность петли по меньшей мере из 4 рибонуклеотидов и первую антисмысловую рибонуклеотидную последовательность по меньшей мере из 20 последовательных рибонуклеотидов, где первая антисмысловая рибонуклеотидная последовательность гибридизуется с первой смысловой рибонуклеотидной последовательностью в молекуле РНК, где первая антисмысловая рибонуклеотидная последовательность способна к гибридизации с первой областью молекулы РНК-мишени, где второй РНК-компонент ковалентно связан через соединительную рибонуклеотидную последовательность, если таковая присутствует, или напрямую, если соединительная рибонуклеотидная последовательность отсутствует, с первым 5'-рибонуклеотидом или первым 3'-рибонуклеотидом, где второй РНК-компонент состоит, в порядке от 5' к 3', из второго 5'-рибонуклеотида, второй последовательности РНК и второго 3'-рибонуклеотида, где вторые 5' и 3'-рибонуклеотиды спариваются друг с другом в молекуле РНК, где вторая последовательность РНК включает вторую смысловую рибонуклеотидную последовательность, вторую последовательность петли по меньшей мере из 4 рибонуклеотидов и вторую антисмысловую рибонуклеотидную последовательность, где вторая смысловая рибонуклеотидная последовательность гибридизуется со второй антисмысловой рибонуклеотидной последовательностью в молекуле РНК, где 5'-лидерная последовательность, если таковая присутствует, состоит из последовательности рибонуклеотидов, ковалентно связанной с первым 5'-рибонуклеотидом, если второй РНК-компонент связан с первым 3'-рибонуклеотидом, или со вторым 5'-рибонуклеотидом, если второй РНК-компонент связан с первым 5'-рибонуклеотидом, и где 3'-трейлерная последовательность, если таковая присутствует, состоит из последовательности рибонуклеотидов, ковалентно связанной со вторым 3'-рибонуклеотидом, если второй РНК-компонент связан с первым 3'-рибонуклеотидом, или с первым 3'-рибонуклеотидом, если второй РНК-компонент связан с первым 5'-рибонуклеотидом. Изобретение позволяет эффективно осуществлять способ даунрегуляции уровня и/или активности молекулы РНК-мишени в организме путем доставки в организм молекулы РНК согласно изобретению. 14 н. и 78 з.п. ф-лы, 37 ил., 1 табл., 20 пр.
1. Молекула РНК для даунрегуляции уровня и/или активности молекулы РНК-мишени, где молекула РНК включает первый РНК-компонент, второй РНК-компонент, который ковалентно связан с первым РНК-компонентом, и, необязательно, одно или более, или все из: (i) соединительной рибонуклеотидной последовательности, которая ковалентно связывает первый и второй РНК-компоненты, (ii) 5'-лидерной последовательности и (iii) 3'-трейлерной последовательности,
где первый РНК-компонент состоит, в порядке от 5' к 3', из первого 5'-рибонуклеотида, первой последовательности РНК и первого 3'-рибонуклеотида, где первые 5' и 3'-рибонуклеотиды спариваются друг с другом в первом РНК-компоненте, где первая последовательность РНК включает первую смысловую рибонуклеотидную последовательность по меньшей мере из 20 последовательных рибонуклеотидов, первую последовательность петли по меньшей мере из 4 рибонуклеотидов и первую антисмысловую рибонуклеотидную последовательность по меньшей мере из 20 последовательных рибонуклеотидов, где первая антисмысловая рибонуклеотидная последовательность гибридизуется с первой смысловой рибонуклеотидной последовательностью в молекуле РНК, где первая антисмысловая рибонуклеотидная последовательность способна к гибридизации с первой областью молекулы РНК-мишени,
где второй РНК-компонент ковалентно связан через соединительную рибонуклеотидную последовательность, если таковая присутствует, или напрямую, если соединительная рибонуклеотидная последовательность отсутствует, с первым 5'-рибонуклеотидом или первым 3'-рибонуклеотидом,
где второй РНК-компонент состоит, в порядке от 5' к 3', из второго 5'-рибонуклеотида, второй последовательности РНК и второго 3'-рибонуклеотида, где вторые 5' и 3'-рибонуклеотиды спариваются друг с другом в молекуле РНК, где вторая последовательность РНК включает вторую смысловую рибонуклеотидную последовательность, вторую последовательность петли по меньшей мере из 4 рибонуклеотидов и вторую антисмысловую рибонуклеотидную последовательность, где вторая смысловая рибонуклеотидная последовательность гибридизуется со второй антисмысловой рибонуклеотидной последовательностью в молекуле РНК,
где 5'-лидерная последовательность, если таковая присутствует, состоит из последовательности рибонуклеотидов, ковалентно связанной с первым 5'-рибонуклеотидом, если второй РНК-компонент связан с первым 3'-рибонуклеотидом, или со вторым 5'-рибонуклеотидом, если второй РНК-компонент связан с первым 5'-рибонуклеотидом, и
где 3'-трейлерная последовательность, если таковая присутствует, состоит из последовательности рибонуклеотидов, ковалентно связанной со вторым 3'-рибонуклеотидом, если второй РНК-компонент связан с первым 3'-рибонуклеотидом, или с первым 3'-рибонуклеотидом, если второй РНК-компонент связан с первым 5'-рибонуклеотидом.
2. Молекула РНК по п.1, где молекула РНК-мишени находится в клетке членистоногого, растения или насекомого.
3. Молекула РНК по п.1 или 2, где по меньшей мере все 20 последовательных рибонуклеотидов первой антисмысловой рибонуклеотидной последовательности способны спариваться с нуклеотидами первой области молекулы РНК-мишени.
4. Молекула РНК по любому из пп.1-3, где первая смысловая рибонуклеотидная последовательность ковалентно связана с первым 5'-рибонуклеотидом без каких-либо промежуточных нуклеотидов, и/или первая антисмысловая рибонуклеотидная последовательность ковалентно связана с первым 3'-рибонуклеотидом без каких-либо промежуточных нуклеотидов.
5. Молекула РНК по любому из пп.1-4, включающая соединительную рибонуклеотидную последовательность, где соединительная рибонуклеотидная последовательность содержит меньше 20 рибонуклеотидов.
6. Молекула РНК по п.5, где соединительная рибонуклеотидная последовательность гибридизуется с молекулой РНК-мишени.
7. Молекула РНК по п.5 или 6, где соединительная рибонуклеотидная последовательность идентична части комплемента молекулы РНК-мишени.
8. Молекула РНК по любому из пп.5-7, где соединительная рибонуклеотидная последовательность имеет длину от 1 до 10 рибонуклеотидов.
9. Молекула РНК по любому из пп.1-8, включающая две или больше смысловых рибонуклеотидных последовательностей, и антисмысловые рибонуклеотидные последовательности, полностью спаренные с ними, которые идентичны по последовательности области молекулы РНК-мишени.
10. Молекула РНК по п.9, где первая и вторая смысловая рибонуклеотидная последовательность идентична по последовательности разным областям одной и той же молекулы РНК-мишени.
11. Молекула РНК по п.9, где первая и вторая смысловая рибонуклеотидная последовательность идентична по последовательности области разных молекул РНК-мишеней.
12. Молекула РНК по любому из пп.1-11, где первая и вторая смысловая рибонуклеотидная последовательность не имеет никаких промежуточных последовательностей петель.
13. Молекула РНК по любому из пп.1-12, где первая и вторая антисмысловая рибонуклеотидная последовательность полностью спарена с первой и второй смысловой рибонуклеотидной последовательностю, соответственно, и каждая из первой и второй антисмысловой рибонуклеотидной последовательности комплементарна области молекулы РНК-мишени.
14. Молекула РНК по любому из пп.1-12, где первая и вторая антисмысловая рибонуклеотидная последовательность полностью спарена с первой и второй смысловой рибонуклеотидной последовательностю, соответственно, и первая и вторая антисмысловая рибонуклеотидная последовательность комплементарна разным областям одной и той же молекулы РНК-мишени.
15. Молекула РНК по любому из пп.1-12, где первая и вторая антисмысловая рибонуклеотидная последовательность полностью спарена с первой и второй смысловой рибонуклеотидной последовательностю, соответственно, и вторая антисмысловая рибонуклеотидная последовательность комплементарна области другой молекулы РНК-мишени, нежели первая антисмысловая рибонуклеотидная последовательность.
16. Молекула РНК по любому из пп.1-15, где первая и вторая смысловая рибонуклеотидная последовательность не содержит никаких промежуточных последовательностей петель.
17. Молекула РНК по любому из пп.1-16, которая представляет собой одиночную цепь рибонуклеотидов, имеющую 5'-конец, по меньшей мере одну смысловую рибонуклеотидную последовательность, которая имеет длину по меньшей мере 21 нуклеотид, антисмысловую рибонуклеотидную последовательность, которая полностью спарена с каждым нуклеотидом смысловой рибонуклеотидной последовательности на протяжении по меньшей мере 21 последовательного нуклеотида, по меньшей мере две последовательности петель и 3'-конец.
18. Молекула РНК по любому из пп.1-16, которая представляет собой одиночную цепь рибонуклеотидов, имеющую 5'-конец, по меньшей мере одну смысловую рибонуклеотидную последовательность, которая имеет длину по меньшей мере 40 нуклеотид, антисмысловую рибонуклеотидную последовательность, которая полностью спарена с каждым нуклеотидом смысловой рибонуклеотидной последовательности на протяжении по меньшей мере 40 последовательного нуклеотида, по меньшей мере две последовательности петель и 3'-конец.
19. Молекула РНК по любому из пп.1-17, которая представляет собой одиночную цепь рибонуклеотидов, включающую 5'-конец, первый РНК-компонент, включающий первую смысловую рибонуклеотидную последовательность, которая имеет длину по меньшей мере 21 нуклеотид, по меньшей мере одну последовательностью петли, первую антисмысловую рибонуклеотидную последовательность, гибридизующуюся с первой смысловой рибонуклеотидной последовательностью на протяжении по меньшей мере 21 последовательного нуклеотида, и второй РНК-компонент, включающий вторую смысловую рибонуклеотидную последовательность, которая имеет длину по меньшей мере 21 нуклеотид, последовательность петли, вторую антисмысловую рибонуклеотидную последовательность, гибридизующуюся со второй смысловой рибонуклеотидной последовательностью на протяжении по меньшей мере 21 последовательного нуклеотида, и 3'-конец, где молекула РНК имеет только один 5'-конец и только один 3'-конец.
20. Молекула РНК по п.19, где рибонуклеотид на 5'-конце и рибонуклеотид на 3'-конце примыкают друг к другу, причем каждый из них спарен и не связан напрямую ковалентной связью.
21. Молекула РНК по любому из пп.1-20, где первая антисмысловая рибонуклеотидная последовательность гибридизуется с первой областью РНК-мишени, вторая антисмысловая рибонуклеотидная последовательность гибридизуется со второй областью РНК-мишени, причем вторая область РНК-мишени отличается от первой области РНК-мишени, и первая и вторая смысловая рибонуклеотидная последовательность примыкают друг к другу, где две антисмысловых последовательности не примыкают друг к другу в молекуле РНК.
22. Молекула РНК по любому из пп.1-20, где первая смысловая рибонуклеотидная последовательность по меньшей мере на 60% идентична первой области РНК-мишени, вторая смысловая рибонуклеотидная последовательность по меньшей мере на 60% идентична второй области РНК-мишени, причем вторая область РНК-мишени отличается от первой области РНК-мишени, и первая и вторая антисмысловая рибонуклеотидная последовательность примыкают друг к другу и гибридизуются с РНК-мишенью, где две смысловых последовательности не примыкают друг к другу в молекуле РНК.
23. Молекула РНК по любому из пп.1-22, имеющая 5'-лидерную последовательность.
24. Молекула РНК по любому из пп.1-19, имеющая 3'-трейлерную последовательность.
25. Молекула РНК по любому из пп.1-12, где каждый рибонуклеотид ковалентно связан с двумя другими нуклеотидами, где молекула РНК представляет собой кольцевую молекулу РНК.
26. Молекула РНК по п.25, где по меньшей мере одна или все последовательности петель в молекуле РНК имеют длину больше 20 нуклеотидов.
27. Молекула РНК по любому из пп.1-14, где молекулы РНК не имеют ни одного или имеют одно или два, или больше выпетливаний, или двухцепочечная область молекулы РНК включает один или два, или больше нуклеотидов, которые не спарены в двухцепочечной области.
28. Молекула РНК по любому из пп.1-27, которая имеет три, четыре или больше петель.
29. Молекула РНК по любому из пп.1-27, которая имеет только две петли.
30. Молекула РНК по любому из пп.1-29, где молекула РНК-мишень находится в эукариотической клетке.
31. Молекула РНК по п.30, где клетка является клеткой растения, клеткой животного или клеткой гриба.
32. Молекула РНК по п.30 или 31, которая присутствует в клетке.
33. Молекула РНК по п.32, которая продуцируется в результате транскрипции в клетке.
34. Молекула РНК по любому из пп.1-25 или 27-33, где по меньшей мере одна из петель имеет длину от 4 до 1000 рибонуклеотидов или от 4 до 200 рибонуклеотидов.
35. Молекула РНК по п.34, где все петли имеют длину от 4 до 1000 рибонуклеотидов или от 4 до 200 рибонуклеотидов.
36. Молекула РНК по п.35, где все петли имеют длину от 4 до 50 рибонуклеотидов.
37. Молекула РНК по любому из пп.30-36, где эукариотическая клетка является клеткой позвоночного, и каждая петля имеет длину от 20 до 30 рибонуклеотидов.
38. Молекула РНК по любому из пп.1-37, где РНК-мишень кодирует белок.
39. Молекула РНК по любому из пп. 1-38, включающая область двухцепочечной РНК (дцРНК), включающую первую смысловую рибонуклеотидную последовательность и первую антисмысловую рибонуклеотидную последовательность, которые способны к гибридизации друг с другом с образованием области дцРНК, где от 5% до 40% рибонуклеотидов области дцРНК спарены с образованием неканонической пары оснований.
40. Молекула РНК по п.39, где от 15% до 30% или от 16% до 25% рибонуклеотидов области дцРНК спарены с образованием неканонической пары оснований.
41. Молекула РНК по п.39 или 40, где по меньшей мере 50%, по меньшей мере 60%, по меньшей мере 70%, по меньшей мере 80%, по меньшей мере 90%, по меньшей мере 95%, по меньшей мере 97% или 100% неканонических пар оснований являются парами оснований G:U.
42. Молекула РНК по любому из пп.39-41, где все из рибонуклеотидов в области дцРНК являются спаренными.
43. Молекула РНК по любому из пп.39-42, где каждый четвертый - каждый шестой рибонуклеотид в области дцРНК образует неканоническую пару оснований.
44. Молекула РНК по любому из пп.39-43, где область дцРНК не включает 8 последовательных канонических пар оснований.
45. Молекула РНК по любому из пп.39-43, где область дцРНК включает по меньшей мере 8 последовательных канонических пар оснований.
46. Молекула РНК по любому из пп.39-45, где все рибонуклеотиды в области дцРНК спарены с образованием канонической пары оснований или неканонической пары оснований, где все из неканонических пар оснований представляют собой пары оснований G:U.
47. Молекула РНК по п.39, где один или более рибонуклеотидов смысловой рибонуклеотидной последовательности и/или один или более рибонуклеотидов антисмысловой рибонуклеотидной последовательности не спарены.
48. Молекула РНК по любому из пп.39-47, где антисмысловая последовательность РНК меньше чем на 100% идентична или идентична на 80-99,9%, или идентична на 90-98%, или идентична на 95-98% по последовательности комплементу области молекулы РНК-мишени.
49. Молекула РНК по любому из пп.39-48, где антисмысловая последовательность РНК на 100% идентична по последовательности области молекулы РНК-мишени.
50. Молекула РНК по любому из пп.39-49, где смысловая и/или антисмысловая рибонуклеотидная последовательность имеет длину по меньшей мере 50, по меньшей мере 100, по меньшей мере 200, по меньшей мере 300, по меньшей мере 400, по меньшей мере 500, по меньшей мере 600, по меньшей мере 700, по меньшей мере 800, по меньшей мере 900, по меньшей мере 1000 или от 100 до 1000 нуклеотидов.
51. Молекула РНК по любому из пп.39-50, где количество рибонуклеотидов в смысловой рибонуклеотидной последовательности составляет от 90% до 110% от количества рибонуклеотидов в антисмысловой рибонуклеотидной последовательности.
52. Молекула РНК по любому из пп.39-51, где количество рибонуклеотидов в смысловой рибонуклеотидной последовательностей равно количеству рибонуклеотидов в антисмысловой рибонуклеотидной последовательности.
53. Молекула РНК по любому из пп.39-52, где первый 3'-рибонуклеотид и второй 5'-рибонуклеотид ковалентно соединены последовательностью петли, состоящей по меньшей мере из 4 рибонуклеотидов или 4-1000 рибонуклеотидов, или 4-200 рибонуклеотидов, 4-50 рибонуклеотидов.
54. Молекула РНК по любому из пп.39-53, где молекула РНК дополнительно включает 5'-удлиняющую последовательность, которая ковалентно связана с первым 5'-рибонуклеотидом, и/или 3'-удлиняющую последовательность, которая ковалентно связана со вторым 3'-рибонуклеотидом.
55. Молекула РНК по любому из пп.39-54, где молекула РНК дополнительно включает 5'-удлиняющую последовательность, которая ковалентно связана со вторым 5'-рибонуклеотидом, и/или 3'-удлиняющую последовательность, которая ковалентно связана с первым 3'-рибонуклеотидом.
56. Молекула РНК по любому из пп.39-55, включающая две или больше области дцРНК, которые являются одинаковыми или разными.
57. Молекула РНК по любому из пп.39-56, где при экспрессии в эукариотической клетке образуется больше молекул асРНК, которые имеют длину 22 и/или 20 рибонуклеотидов, по сравнению с процессингом аналогичной молекулы РНК, имеющей соответствующую область дцРНК, которая полностью спарена с образованием канонических пар оснований.
58. Молекула РНК по любому из пп.39-57, которая включает один или более дополнительных признаков, как определено в любом одном или более пп.1-38.
59. Выделенный полинуклеотид для даунрегуляции уровня и/или активности молекулы РНК-мишени, где полинуклеотид кодирует молекулу РНК по любому из пп.1-58.
60. Полинуклеотид по п.59, который является конструкцией ДНК.
61. Полинуклеотид по п.59 или 60, функционально связанный с промотором, способным направлять экспрессию молекулы РНК в клетке-хозяине.
62. Полинуклеотид по п.61, где промотор является промотором РНК-полимеразы, таким как промотор РНК-полимеразы III, промотор РНК-полимеразы II, или промотором, который функционирует in vitro.
63. Полинуклеотид по любому из пп.59-62, который кодирует молекулу РНК-предшественника, включающую интрон по меньшей мере в одной последовательности петли, который может быть удален при сплайсинге в ходе транскрипции полинуклеотида в клетке-хозяине.
64. Вектор для экспрессии молекулы РНК по любому из пп. 1-58, включающий полинуклеотид по любому из пп.59-63.
65. Вектор по п.64, который является вирусным вектором.
66. Клетка-хозяин для экспрессии молекулы РНК по любому из пп. 1-58, включающая полинуклеотид по любому из пп.59-63.
67. Клетка-хозяин по п.66, которая является клеткой бактерии, клеткой гриба, клеткой растения или клеткой животного.
68. Клетка-хозяин по п.66 или 67, которая является мертвой.
69. Полинуклеотид по п.61 или 62 или клетка-хозяин по любому из пп.66-68, кодирующие и/или включающие молекулу РНК по любому из пп.39-58, где промоторная область полинуклеотида имеет более низкий уровень метилирования, такой как меньше чем 50%, меньше чем 40%, меньше чем 30% или меньше чем 20% по сравнению с промотором соответствующего полинуклеотида, кодирующего молекулу РНК, имеющую соответствующую область дцРНК, которая полностью спарена с образованием канонических пар оснований.
70. Клетка-хозяин по любому из пп.66-69, включающая по меньшей мере две копии полинуклеотида, кодирующего молекулу РНК по любому из пп.39-58, и где:
i) уровень снижения экспрессии или активности молекулы РНК-мишени в эукариотической клетке является, по меньшей мере, таким же по сравнению с таким уровнем, если клетка содержала одну копию полинуклеотида, и/или
ii) уровень снижения экспрессии или активности молекулы РНК-мишени в эукариотической клетке является более низким по сравнению с соответствующей клеткой, включающей молекулу РНК, содержащую соответствующую область дцРНК, которая полностью спарена с образованием канонических пар оснований.
71. Клетка-хозяин по любому из пп.66-70, где клетка кодирует и/или включает молекулу РНК по любому из пп.39-58, и уровень смысловой рибонуклеотидной последовательности в клетке составляет меньше 50% от уровня антисмыслового рибонуклеотида.
72. Растение для получения молекулы дцРНК, включающее одно или более, или все из молекулы РНК по любому из пп.1-58, полинуклеотида по любому из пп.59-63 или 69, вектора по п.64 или по 65 или клетки-хозяина по любому из пп.66-71.
73. Растение по п.72, который является трансгенным растением, включающим полинуклеотид по любому из пп.59-63 или 69.
74. Растение по п.73, где полинуклеотид стабильно интегрирован в геном организма.
75. Способ получения молекулы РНК по любому из пп.1-58, включающий экспрессию полинуклеотида по любому из пп.59-63 или 69 в клетке-хозяине или бесклеточной системе экспрессии.
76. Способ по п.75, дополнительно включающий, по меньшей мере, частичную очистку молекулы РНК.
77. Способ получения растения по п.73 или 74, включающий введение полинуклеотида по любому из пп.59-63 или 69 в клетку таким образом, чтобы он стабильно интегрировался в геном клетки, и создание растения из клетки.
78. Экстракт клетки-хозяина по любому из пп.66-71 для даунрегуляции уровня и/или активности молекулы РНК-мишени, где экстракт включает молекулу РНК по любому из пп.1-58, и/или полинуклеотид по любому из пп.59-63 или 69.
79. Композиция для даунрегуляции уровня и/или активности молекулы РНК-мишени, где композиция включает одно или более из молекулы РНК по любому из пп.1-58, полинуклеотида по любому из пп.59-63 или 69, вектора по п.64 или 65, клетки-хозяина по любому из пп.66-71 или экстракта по п.78, и один или более подходящих носителей.
80. Композиция по п.79, которая является фармацевтической композицией.
81. Композиция по п.79, подходящая для применения в поле.
82. Композиция по п.81, где поле включает растения.
83. Композиция по любому из пп.79-82, дополнительно включающая по меньшей мере одно соединение, которое повышает стабильность молекулы РНК, или полинуклеотида, и/или которое способствует захвату молекулы РНК, или полинуклеотида клеткой организма.
84. Композиция по п.83, где соединение является средством, способствующим трансфекции.
85. Способ даунрегуляции уровня и/или активности молекулы РНК-мишени в организме, включающий доставку в организм молекулы РНК по любому из пп.1-58.
86. Способ даунрегуляции уровня и/или активности молекулы РНК-мишени в организме, включающий доставку в организм полинуклеотида по любому из пп. 59-63 или 69.
87. Способ по п.85 или 86, где молекула РНК-мишени кодирует белок.
88. Способ борьбы с растением, включающий доставку в растение молекулы РНК по любому из пп.1-58, где молекула РНК оказывает токсический эффект в отношении растения.
89. Способ борьбы с членистоногим, включающий доставку в членистоногое молекулы РНК по любому из пп. 1-58, где молекула РНК оказывает токсический эффект в отношении членистоногого.
90. Способ по п.89, где членистоногое питается частью растения, содержащей молекулу РНК.
91. Молекула РНК по любому из пп.1-58, полинуклеотид по любому из пп.59-63 или 69, вектор по п.64 или 65, клетка-хозяин по любому из пп.66-71, экстракт по п.78 или композиция по любому из пп.79-84 для применения в лечении заболевания у субъекта, где молекула РНК оказывает благоприятный эффект в отношении по меньшей мере одного симптома заболевания.
92. Членистоногое, содержащее молекулу РНК по любому из пп. 1-58, для борьбы с членистоногим.
| WO 2016100333 A1, 23.06.2016 | |||
| WO 2014107763 A1, 17.07.2014 | |||
| DENNIS MELVIN B., Welfare issues of genetically modified animals, ILAR Journal, 2002, Vol.43, No.2, pp.100-109 | |||
| ZHOU HONGXIA, et al., Developing tTA transgenic rats for inducible and reversible gene expression, International Journal of Biological Sciences, 2009, 5, pp.171-181 | |||
| Мазуренко |
Авторы
Даты
2024-05-28—Публикация
2018-09-17—Подача