Изобретение относится к области медицинской микробиологии, в частности к методам генотипирования возбудителей инфекционных заболеваний, которые используются при молекулярно-генетическом мониторинге возбудителя коклюша с целью выявления эпидемических клонов с измененной генетической структурой и слежения за их распространением на территории России.
Коклюш - высококонтагиозное антропонозное воздушно-капельное инфекционное заболевание. Введение массовой иммунизации детского населения в середине прошлого века коренным образом изменило характер течения эпидемического процесса коклюшной инфекции во всем мире и способствовала значительному уменьшению количества случаев заболеваний, тяжести течения болезни и резкому снижению летальности (Лыткина И.Н. и др., 2004, Петрова М.С.и др., 2012, Краснов В.В., 2018, Бабаченко Н.В. и др., 2019, Нестерова Ю.В. 2019, Государственный доклад 2021, 2022). В настоящее время, несмотря на высокий уровень вакцинации населения, увеличилось количество случаев заболеваемости коклюшем во всех возрастных группах, которая обусловлена целым рядом факторов: снижение иммунитета с возрастом, недостатки вакцинации ацеллюлярными вакцинами, адаптация возбудителя под селективным давлением иммунизации, улучшение диагностики за счет широкого применения молекулярно-генетических методов (Mooi F 2010, Kaffy K. et al., 2017, Skoff Т.Н. et al., 2019, Fullen A.R. 2020, Bouchez V. 2021). Это свидетельствует о продолжающейся широкой циркуляции возбудителя коклюша (Лыткина И.Н. и др., 2004, Мазурова И.К. и др., 2008, Краснов В.В., 2018, Нестерова Ю.В. 2019, Государственный доклад 2021, 2022). Кроме того, в последние годы в структуре заболеваемости увеличивается удельный вес стертых и легких форм болезни среди детей старшей возрастной группы и взрослых (Борисова О.Ю. и др., 2010, Краснов В.В. 2018, Нестерова Ю.В. 2019, Скирда Т.А. и др., 2020, 2021, 2022, Государственный доклад 2021, 2022). Причиной этому, является изменение фенотипических и генотипических свойств возбудителя коклюша под влиянием длительной массовой иммунизации.
Молекулярно-генетический мониторинг штаммов B. pertussis проводится во многих странах мира и в России. К настоящему времени штаммовый SN-полиморфизм (SNP) В. pertussis описан в генах, кодирующих поверхностные белки и в генах, детерминирующих основные факторы адгезии, колонизации и патогенности (Шинкарев А.С 2007, Борисова О.Ю. 2008, 2008, 2012, 2012, 2016, Ивашинникова Г.А. 2012, 2012, 2013, 2013, 2013,). Было установлено, что штаммы с новыми «невакцинными» аллелями этих генов постепенно вытесняют штаммы со старыми «вакцинными» аллелями генов, т.е. происходит селекция штаммов с измененной структурой антигенных детерминант основных факторов патогенности, которые отличаются по антигенной специфичности от структуры вакцинных штаммов, используемых для производства профилактических препаратов. (Шинкарев А.С 2007, Мазурова И.К. 2008, Борисова О.Ю. 2008, 2008, 2012, 2012, 2016, Ивашинникова Г.А. 2012, 2012, 2013, 2013, 2013, Мерцалова Н.У. 2009, Mooi F 2010, Kaffy K. et al., 2017, Moriuchi Т. 2017, Skoff Т.Н. et al., 2019, Carriquiriborde F. 2019, Fullen A.R. 2020, Bouchez V. 2021, Wu S. 2021, Alai S. 2022, Wakimoto Y, 2022).
Одним из методов, используемых для мониторинга за возбудителем коклюша, является мультилокусное антигенное сиквенс-типирование (MAST). Данный метод основан на секвенировании фрагментов основных протективных антигенов возбудителя, что позволяет выявлять генетические различия и оценивать клональный состав циркулирующей популяции В. pertussis.
В настоящее время для изучения особенностей генетической структуры основных факторов патогенности штаммов В. pertussis необходимо выделение чистой бактериальной культуры В. pertussis. Это занимает длительное время культивирования (до 7 дней) при общей эффективности бактериологического исследования до вида 20%, что связано с приемом антибактериальных препаратов, сложностью выделения бактериальной культуры у детей раннего возраста. (Ценева Г.Я. и др., 2003).
Задачей настоящего изобретения является разработка способа генотипирования B. pertussis по схеме MAST2 непосредственно из клинических образцов (мазков с задней стенки ротоглотки) без выделения чистой культуры возбудителя на основе nested-ПЦР для наблюдения за циркулирующим возбудителем коклюша, выявления эпидемических клонов при молекулярно-генетическом мониторинге в системе эпидемиологического надзора за коклюшной инфекцией.
Генотипирование по схеме MAST2 направлено на исследование фрагментов генов ptxP-fim3-prn, кодирующих промоторную ptxP область коклюшного токсина, фимбриальный Fim3 белок и пертактин. Это позволяет идентифицировать гены, кодирующие детерминанты, которые входят в состав бесклеточных коклюшных вакцин (Mooi F.R. 2010). Способ генотипирования из клинических образцов сокращает время получения генотипа до трех дней от момента выделения ДНК.
По данным предыдущих исследований в рамках работы Референс центра по мониторингу возбудителей коклюша и дифтерии в ФБУН «МНИИЭМ им. Г.Н. Габричевского» Роспотребнадзора показано, что в динамике эпидемического процесса коклюшной инфекции на территории России циркулировали штаммы девятнадцати (генотипирование по MAST2) генотипов: 111, 211, 219, 123, 121, 113, 132, 341, 342, 122, 221, 319, 112, 311, 329, 312, 323, 224 и 322, большинство из которых зарегистрированы с конца 1990-х годов (Алешкин В.А. 2013, Борисова О.Ю. 2012, 2016, Ивашинникова Г.А. 2012, 2012, 2013, 2013, 2013).
Ранее нами были предложены способы генотипирования B. pertussis (Патент РФ №2299908, Патент РФ №2495132). Однако, данные способы генотипирования предлагалось проводить после выделения чистой бактериальной культуры возбудителя на плотных питательных средах и занимало до 10-14 дней от момента взятия биологического материала от больного.
За рубежом были представлены исследования по генотипированию возбудителя коклюша с клинического материала методом MLVA-типирования (Moriuchi Т., 2017). Однако для проведения таких исследований необходимо дорогостоящее специализированное оборудование с программным обеспечением и импортные реагенты, кроме того, авторы не предоставляют информацию о возможностях определения генотипа с малым количеством патогенного материала на тампоне.
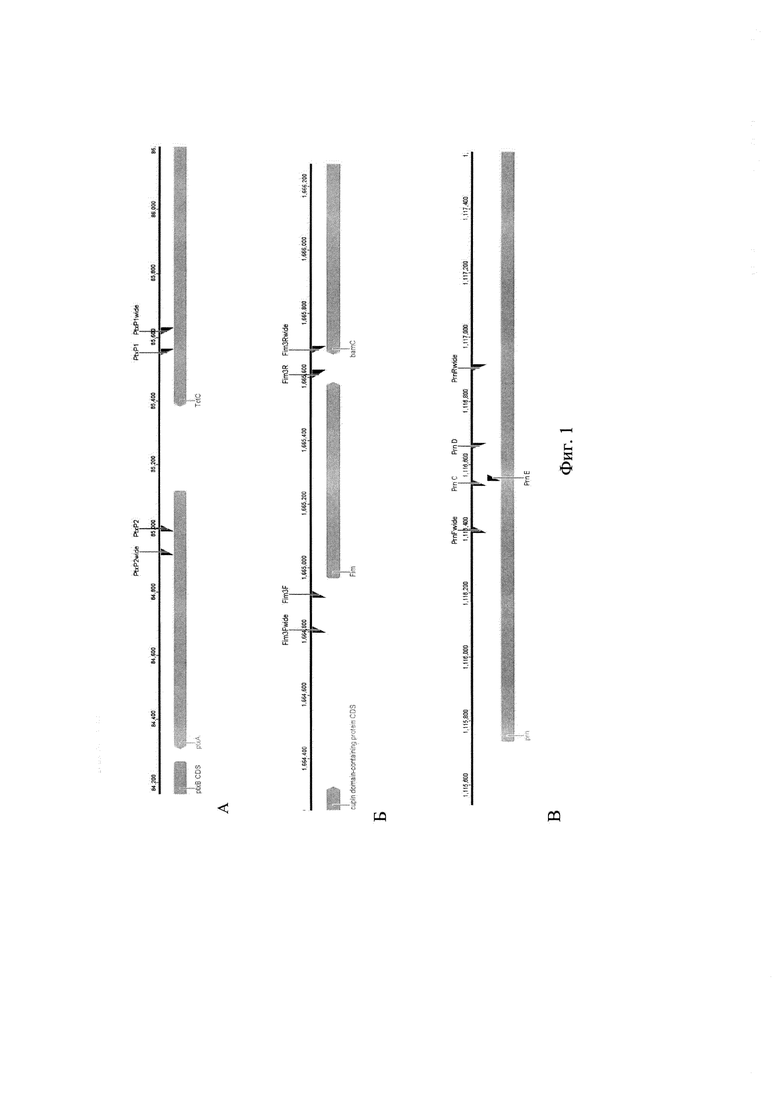
В соответствии с поставленной задачей был разработан способ генотипирования B. pertussis на основе nested-ПЦР по структуре промотора коклюшного токсина (ptxP), белка пертактина (prn) и фимбриального белка Fim3 (fim3), предусматривающий выделение ДНК из клинических образцов (мазков с задней стенки ротоглотки), проведение полимеразной цепной реакции (первого раунда nested-ПЦР) с внешними специфическими праймерами wide F-R для получения ампликонов фрагментов генов промотора коклюшного токсина (ptxP), фимбриального белка Fim3 (fim3) и пертактина (prn). Полученные ПЦР-продукты являются матрицей для проведения второго раунда nested-ПЦР с внутренними праймерами для получения ампликонов фрагментов генов промотора коклюшного токсина (ptxP), фимбриального белка Fim3 (fim3) и пертактина (prn). В результате проведенной nested-ПЦР нарабатывают ПЦР-продукты фрагментов исследуемых генов, которые по результатам мультилокусного антигенного сиквенс-типирования MAST2 можно определить аллельные варианты.
Изобретение поясняется Фиг. 1 и таблицей, где Фиг. 1 - положение праймеров, фланкирующих участки генов, кодирующих промотор коклюшного токсина (ptxP), фимбриальный белок Fim3 (fim3) и белок пертактин (prn). Таб. 1 - праймеры, используемые в nested-ПЦР. Примечание: A - PtxP, Б - Fim3, B - Prn.
Способ осуществляется следующим образом.
Биологический материал (мазок с задней стенки ротоглотки) в 0,5 мл физиологического раствора или транспортной среды для хранения и транспортировки респираторных мазков перемешивали на вортексе и центрифугировали при 5000 g в течение 5 сек. Отжимали и удаляли тампоны, после чего осаждали смывы центрифугированием при 13000 g в течение 2 мин. Далее удаляли супернатант, оставляя в пробирке полученный осадок и 100 мкл надосадочной жидкости, вортексировали и выделяли тотальную ДНК с помощью набора реагентов «Рибо-преп» (ООО «НекстБио», Москва) в соответствии с инструкцией по применению.
Для проведения 1 раунда nested-ПЦР были использованы следующие сконструированные нами праймеры (Фиг. 1, Табл. 1).
Для 2 раунда использовались опубликованные праймеры (Mooi F.R. 2010). Реакционная смесь содержала: 10×ПЦР буфер с (NH4)2SO4, 25 mM MgCI2, 2 mM смесь dNTPs, betaine, по 10 pmol праймеров, 2,5 ед. Taq ДНК-полимеразы, 2 мкл препарата ДНК. Общий объем реакционной смеси 30 мкл. Амплификацию проводили с помощью прибора «Терцик» (ДНК-технология, Россия) при следующих условиях: для ptxP: 95°С 10 мин, 30 циклов 95°С 15 сек, 55°С 30 сек, 72°С 1 мин, и 72°С 10 мин;
для prn: 94°С 10 мин, 40 циклов 94°С 1 мин, 66°С 1 мин 30 сек, 72°С 2 мин;
для fim3: 95°С 15 мин, 30 циклов 95°С 30 сек., 56°С 30 сек, 72°С 1 мин.
Детекцию продуктов амплификации осуществляли методом горизонтального электрофореза в агарозном геле (Хеликон, Россия) в 50× ТАЕ-буфере (Евроген, Россия) в камере SUB-CELL® GT (Bio-Rad Laboratories, США) с применением источника питания Power Supply Model 2002 / 2.0 (Bio-Rad Laboratories, США). Для определения длины амплифицированных фрагментов использовали маркер молекулярных весов ДНК GeneRuler 100 bp DNA Ladder (Евроген, Россия).
ПЦР-продукты (10 мкл) смешивали с 6х DNA Loading Dye (Евроген, Россия) и 10000х SYBR Green I (Люмипроб, Россия), после чего образцы помещали в лунки 1,5% агарозного геля и подвергали действию постоянного электрического тока. Электрофорез проводили при напряжении 160 V в течение 60 мин. Продукты амплификации визуализировали с помощью гельдокументирующей системы Quantum-ST4-1100/26М (Vilber Lourmat, Франция).
Полученные после 2 раунда ПЦР-продукты фрагментов генов ptxP (573 п. н.), fim3 (715 п. н.), prn (526 п. н) подвергались мультилокусному антигенному сиквенс типированию с последующим анализом с помощью базы данных https://bigsdb.pasteur.fr/bordetella/.
Способ был апробирован на 22 положительных образцах, содержащих ДНК В. pertussis.
Пример. Было проверено 22 образцов ДНК В. pertussis, полученных из клинических образцов (мазков с задней стенки ротоглотки), которые были собраны от больных коклюшем детей с клинической симптоматикой заболевания, в возрасте от 1 месяца до 3 лет.Все образцы были охарактеризованы методом мультилокусного антигенного сиквенс-типирования фрагментов трех генов ptxP, fim3 и prn по схеме MAST2. Было установлено, что у 63,6% (14 детей) - присутствует 321 генотип, а у 36,4% (8 детей) - 322 генотип, что свидетельствует о том, что у всех детей обнаружены новые «невакцинные» аллели гена ptxP, кодирующего промотор коклюшного токсина, и prn, кодирующего пертактин, в то время как по структуре гена fim3, кодирующего фибриальный Fim3 белок, у большинства детей выявлен старый «вакцинный» аллель гена.
Проведенные исследования доказывают, что использование разработанного способа генотипирования В. pertussis на основе nested-ПЦР позволяют наработать ПЦР-продукты, которые подвергают MAST-типированию с целью идентификации аллельных вариантов трех генов.
Техническим результатом заявленного изобретения является тот факт, что:
* заявляемый способ генотипирования B. pertussis осуществляется непосредственно из клинического материала при молекулярно-генетическом мониторинге возбудителя коклюша с целью ускоренного выявления эпидемических клонов B. pertussis с измененной структурой гена ptxP промотора коклюшного токсина, гена фимбриального белка Fim3 (fim3) и гена пертактина (prn);
* проведение генотипирования возбудителя коклюша непосредственно из клинического материала позволит оценить генетическую структуру циркулирующей популяции возбудителя в течение трех дней от момента выделения ДНК;
* применение этого способа генотипирования позволит в ускоренные сроки модернизировать существующие вакцинные препараты, которые будут обладать высокой протективной активностью в отношении циркулирующих штаммов возбудителя, вызывающих заболевание коклюшем;
* предлагаемый способ генотипирования является отечественным инструментом для проведения эпидемиологического надзора за биологическими рисками, в частности, за возбудителем коклюша и распространением коклюшной инфекции на территории Российской Федерации.
Литература
1. Алешкин В.А., Борисова О.Ю., Алешкин А.В., Ивашинникова Г.А., Мазурова И.К., Кафарская Л.И., Донских Е.Е., Пименова А.С.Клональный состав популяции штаммов Bordetella pertussis, выделенных в России. // Инфекционные болезни. - 2013. - том 11, №1. - С. 103-109.
2. Бабаченко И.В., Нестерова Ю.В., Чернышова Ю.Ю., Карасев В.В., Починяева Л.Н., Калисникова Е.П. Клинико-эпидемиологические аспекты коклюша у детей в условиях массовой иммунизации // Журнал инфектологии. - 2019. - №11(2). - С. 88-96.
3. Борисова О.Ю., Комбарова С.Ю., Гадуа Н.Т., Мазурова И.К. Молекулярно-генетический метод ускоренного выявления штаммов Bordetella pertussis, обладающих различными ptx генами // Журнал микробиологии, эпидемиологии и иммунобиологии. - 2008. - №5. - С. 80-83.
4. Борисова О.Ю., Комбарова С.Ю., Гадуа Н.Т., Мерцалова Н.У., Шинкарев А.С, Захарова Н.С., Пяева А.П., Лыткина И.Н., Требунских И.П., Салова Н.Я., Савинкова B.C., Скачкова В.Г., Мазурова И.К. Молекулярно-генетический мониторинг штаммов Bordetella pertussis, выделенных в различные периоды эпидемического процесса коклюшной инфекции // Материалы четвертой международной конференции «Идеи Пастера в борьбе с инфекциями», 2-4 июня 2008 г., Санкт - Петербург.- 2008. - С. 7.
5. Борисова О.Ю., Петрова М.С., Мазурова И.К., Лыткина И.Н., Попова О.П., Гадуа Н.Т., Мерцалова Н.У., Захарова Н.С., Пяева А.П., Салова Н.Я., Требунских И.П., Комбарова С.Ю, Шинкарев А.С., Скачкова В.Г., Савинкова B.C., Алёшкин В.А. Особенности коклюшной инфекции в различные периоды эпидемического процесса в г. Москве. // Журнал эпидемиология и вакцинопрофилактика. - 2010. - №4. - С. 33-39.
6. Борисова О.Ю., Мазурова И.К., Ивашинникова Г.А., Гадуа Н.Т., Рудакова И.А., Салова Н.Я., Абасова Ф.М., Требунских И.П., Наретя Н.Д., Алексеева Л.А., Якунина О.Ю., Mooi F., Алешкин В.А. Генетическая характеристика штаммов Bordetella pertussis, выделенных от больных коклюшем в России. // Медицинский альманах. - 2012. - №2 (21). - С. 30-34.
7. Борисова О.Ю., Мазурова И.К., Ивашинникова Г.А., Захарова Н.С., Мерцалова Н.У., Зайцев Е.М., Салова Н.Я., Абасова Ф.М., Требунских И.П., Скачкова В.Г., Панферова Р.А., Алексеева Л.А., Якунина О.Ю., Наретя Н.Д., van Gent М., Mooi F., Алешкин В.А. Особенности распространения штаммов B. pertussis, выделенных от больных коклюшем, с различными аллельными вариантами гена, кодирующего промоторную область коклюшного токсина (ptxP). // Эпидемиология и инфекционные болезни. Актуальные вопросы. - 2012. - №2. - С. 14-19.
8. Борисова О.Ю., Мазурова И.К., Ивашинникова Г.А., Гадуа Н.Т., Рудакова И.А., Салова Н.Я., Требунских И.П., Скачкова В.Г., Панферова Р.А., Mooi F., Алешкин В.А. Характеристика штаммов Bordetella pertussis, выделенных от больных коклюшем в г. Москве, с помощью мультилокусного секвенирования. // Журнал микробиологии, эпидемиологии и иммунобиологии. - 2012. - №2. - С. 28-34.
9. Борисова О.Ю., Гадуа Н.Т., Пименова А.С., Петрова М.С., Попова О.П., Алешкин В.А., Кафарская Л.И., Донских Е.Е., Юсуф Е.В., Остапенко Н.А., Москвина Т.И., Т.А. Щербакова. Структура популяции штаммов возбудителя коклюша на территории России. // Эпидемиология и Вакцинопрофилактика. - 2016. - №4 (89). - С. 22-28.
10. Ивашинникова Г.А., Борисова О.Ю., Гадуа Н.Т., Рудакова И.А., Мазурова И.К. Применение метода мультилокусного секвенирования для наблюдения за циркулирующими штаммами B. pertussis. // Здоровье населения и среда обитания. - 2012. - №9. - С. 28-30.
11. Ивашинникова Г.А., Борисова О.Ю., Алешкин А.В., Пименова А.С.Ускоренный молекулярно-генетический метод типирования штаммов Bordetella pertussis по структуре промотора РТХР коклюшного токсина. // Астраханский медицинский журнал. - 2012. - №7 (4). - С. 121-124.
12. Ивашинникова Г.А., Борисова О.Ю., Пименова А.С. Ускоренный метод мониторинга штаммов Bordetella pertussis по структуре промотора коклюшного токсина ptxP. // Материалы V Ежегодного Всероссийского Конгресса по инфекционным болезням. - 2013. - С. 169.
13. Ивашинникова Г.А., Борисова О.Ю., Алёшкин А.В., Пименова A.С., Алешкин В.А. Совершенствование генотипирования штаммов B. pertussis. // Детские инфекции. - 2013. - №1. - С. 34-36.
14. Ивашинникова Г.А. Диссертация на соискание кандидата биологиченских наук «Молекулярно-генетическая характеристика штаммов B. pertussis и соверешенствование микробиологического мониторинга коклюшной инфекции». - 2013.
15. Краснов В.В. Коклюш у детей первого года жизни / И.И. Краснов, К.Ф. Ильяненков, Л.Р. Павлович, М.В. Кузмичева // Детские инфекции. - 2018. - Т. 1, №1. - С. 12-17.
16. Лыткина И.Н., Чистякова Г.Г., Н.Н. Филатов. Заболеваемость коклюшем в Москве и организация мероприятий по ее снижению. // Новости вакцинопрофилактики: Вакцинация. - 2004. - №35. - С. 8-9.
17. Мазурова И.К., Борисова О.Ю., Комбарова С.Ю., Гадуа Н.Т., Алешкин В.А. Динамика изменчивости основных генов патогенности штаммов Bordetella pertussis, выделенных от больных коклюшем в г. Москве (1948 - 2005 гг.) // Журнал молекулярной медицины. - 2008. - №1. - С. 40-45.
18. Мерцалова Н.У., Борисова О.Ю., Шинкарев А.С., Брицина М.В., Озерецковская М.Н., Мазурова И.К., Алешкин В.А., Гадуа Н.Т., Захарова Н.С.Динамика изменений патогенных свойств штаммов Bordetella pertussis. // Журн. Микробиол. - 2009. - №6. - стр. 7-11.
19. Нестерова Ю.В. Клинико-диагностическое значение генетических маркеров Bordetella pertussis у контактных лиц в семейных очагах / Ю.В. Нестерова, А.Ю. Медкова, И.В. Бабаченко и др. // Журнал инфектологии. - 2019. - Т. 11, №1. - С. 17-24.
20. О состоянии санитарно-эпидемиологического благополучия населения в Российской Федерации в 2021 году: Государственный доклад / Федеральная служба по надзору в сфере защиты прав потребителей и благополучия человека. - М: Федеральная служба по надзору в сфере защиты прав потребителей и благополучия человека, 2022.
21. О состоянии санитарно-эпидемиологического благополучия населения в Российской Федерации в 2022 году: Государственный доклад / Федеральная служба по надзору в сфере защиты прав потребителей и благополучия человека. - М: Федеральная служба по надзору в сфере защиты прав потребителей и благополучия человека, 2023.
22. Патент на изобретение «Способ дифференциации штаммов Bordetella pertussis» №2299908, зарегистрированный в Государственном реестре изобретений Российской Федерации 27 мая 2007 г. (Борисова О.Ю., Комбарова С.Ю., Алешкин В.А., Мазурова И.К.).
23. Патент 2495132 Российская Федерация, МПК C12Q 1/68 С12Р 19/30, C12N 1/20 C12R 1/01. Способ молекулярно-генетического типирования штаммов Bordetella pertussis / Борисова О.Ю., Ивашинникова Г.А., Гадуа Н.Т., Мазурова И.К., Алешкин В.А. заявитель и патентообладатель: ФБУН МНИИЭМ им. Г.Н. Габричевского Роспотребнадзора (RU). - №2012141063/10; заявл. 26.09.2012; опубл. 10.10.2013, Бюл. №28. - 10 с.
24. Петрова М.С, Попова О.П., Борисова О.Ю., Абрамова Е.Н., Вартанян Р.В., Келли Е.И. Коклюш у детей раннего возраста. // Эпидемиология и инфекционные болезни. - 2012. - №6. - С. 12-24.
25. Скирда Т.А., Борисова О.Ю., Петрова М.С., Борисова А.Б., Комбарова С.Ю. Серологическая диагностика коклюша у лиц старшего возраста. // Клиническая лабораторная диагностика. - 2020. - 65(8). - С. 492-495.
26. Скирда, Т.А. Противококлюшные антитела классов IgM, IgG и IgA у беременных в I-II-III триместрах / Т.А. Скирда, О.Ю. Борисова, Н.Т. Гадуа, А.С.Пименова, И.И. Механтьев, Ю.И. Степкин, И.М. Дегтярева, С. В. Бредихин, М.С.Петрова, А.Б. Борисова, А.В. Сафронова, С.Ю. Комбарова. // Журнал инфектологии. - 2021. - Т. 13, №3. - С. 30-38.
27. Скирда, Т.А. Определение противококлюшных антител у школьников с длительным кашлем / Т.А. Скирда, О.Ю. Борисова, А.Б. Борисова, С.Ю. Комбарова, А.С.Пименова, Н.Т. Гадуа, И.А. Чагина, М.С.Петрова, Л.И. Кафарская // Журнал инфектологии. - 2023. - Том 15, №1. - С. 93-100.
28. Ценева Г.Я., Курова Н.Н. Микробиологическая характеристика возбудителя коклюша и лабораторная диагностика коклюша // Клиническая микробиология и антимикробная химиотерапия. - 2003. - №4 (5). - С. 329-341.
29. Шинкарев А.С., Мерцалова Н.У., Мазурова И.К., Борисова О.Ю., Захарова Н.С., Озерецковская М.Н., Зайцев Е.М., Поддубиков А.В., Брицина М.В., Бажанова И.Г. Современные штаммы Bordetella pertussis: молекулярная и иммунобиологическая характеристика // Журнал эпидемиологии, микробиологии и иммунобиологии. - 2007. - №4. - С. 20-25.
30. Alai S., Gautam M., Palkar S., Oswal J., Gairola S., Dhotre D.P. Characterization of Bordetella pertussis Strains Isolated from India. // Pathogens. - 2022. - 11(7). - P. 794.
31. Bouchez V., Guillot S., Landier A., Armatys N., Matczak S. et al. Evolution of Bordetella pertussis over a 23-year period in France, 1996 to 2018. // Euro Surveill. - 2021. - 26(37). - P. 2001213.
32. Carriquiriborde F., Regidor V., Aispuro P.M., Magali G., Bartel E., Bottero D., Hozbor D. Rare Detection of Bordetella pertussis Pertactin-Deficient Strains in Argentina. // Emerging Infectious Diseases. - 2019. - 25 (11). - P. 2048-2054.
33. Fullen A.R., Yount K.S., Dubey P., Deora R. Whoop! There it is: The surprising resurgence of pertussis. // PLoS Pathogens. - 2020. - 16 (7). - P. 1-6.
34. Kaffy K., Guiso N., Diawara I., Zerouali K., Slaoui В., Jouhadi Z., Zineddine A., Belabbes H., Elmdaghri N. Epidemiology of pertussis in Casablanca (Marocco): contribution of conventional and molecular diagnosis tools. // BMC Infectious Diseases. - 2017. - 17(1). - P. 348.
35. Moriuchi Т., Vichit O., Yong Vutthikol Y., Md Shafiqul Hossain S., Samnang C., Toda K., Grabovac V., Hiramatsu Y., OtsukaN., Shibayama K, Kamachi K. Molecular epidemiology of Bordetella pertussis in Cambodia determined by direct genotyping of clinical specimens. // Int J Infect Dis. - 2017. - 62. - P. 56-58.
36. Mooi F.R. Bordetella pertussis and vaccination: the persistence of a genetically monomorphic pathogen. // J. Infect Genet Evol. - 2010. - Vol.10(1). - P. 36-49.
37. Skoff Т.Н., Hadler S., Hariri S. The epidemiology of nationally reported pertussis in the United States, 2000-2016. // Clinical Infectious Diseases. - 2019. - Vol.68, №10. - P. 1634-1640.
38. Wakimoto Y., Otsuka N., Yanagawa Y., Koide K., Kamachi K., Shibayama K., Teruya K., Kikuchi Y., Oka S. The First Reported Case of Bordetella pertussis Bacteremia in a Patient With Human Immunodeficiency Virus Infection. // Open Forum Infect Dis. - 2022. - 9(3).
39. Wu S., Hu Q., Yang C, Zhou H., Chen H., Zhang Y., Jiang M., He Y., Shi X. Molecular epidemiology of Bordetella pertussis and analysis of vaccine antigen genes from clinical isolates from Shenzhen, China. // Annals of Clinical Microbiology and Antimicrobials volume. - 2021. - 20(1). - P. 53.
--->
<?xml version="1.0" encoding="ISO-8859-1"?>
<!DOCTYPE ST26SequenceListing SYSTEM "ST26SequenceListing_V1_3.dtd" PUBLIC "-//WIPO//DTD Sequence Listing 1.3//EN">
<ST26SequenceListing productionDate="2023-11-20" softwareVersion="2.3.0" softwareName="WIPO Sequence" fileName="PCR_primers. 20.11.23.xml" dtdVersion="V1_3">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2023125556/20 (056428)</ApplicationNumberText>
<FilingDate>2023-10-05</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>2023125556/20 (056428)</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное Бюджетное учреждение науки "Московский научно-исследовательский институт эпидемиологии и микробиологии им. Г.Н. Габричевского" Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека</ApplicantName>
<ApplicantNameLatin>Federal'noe Byudzhetnoe uchrezhdenie nauki " Moskovskij nauchno-issledovatel'skij institut epidemiologii i mikrobiologii im.G.N.Gabrichevskogo" Federal'noj sluzhby po nadzoru v sfere zashchity prav potrebitelej i blagopoluchiya cheloveka</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ генотипирования B.pertussis из клинических образцов на основе вложенной ПЦР </InventionTitle>
<SequenceTotalQuantity>13</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bordetella pertussis</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atatcccgaattcgtcgcct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bordetella pertussis</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagcacattgtcgttgtttcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bordetella pertussis</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>catgctaatggcctccggt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bordetella pertussis</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcgtgctgaaggacaagatgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bordetella pertussis</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggcggtgtctgtgttggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bordetella pertussis</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtcagcgtcagcttcac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bordetella pertussis</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aatcgtcctgctcaaccgcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bordetella pertussis</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtatacggtggcgggagga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bordetella pertussis</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gacctgatattctgatgccg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bordetella pertussis</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgcaaggcttgccggttttttttgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>17</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bordetella pertussis</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtgcggttcccggcggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bordetella pertussis</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgcggttcccggcggcttc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bordetella pertussis</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctccacgctggagcccg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБ МОЛЕКУЛЯРНО-ГЕНЕТИЧЕСКОГО ТИПИРОВАНИЯ ШТАММОВ Bordetella pertussis | 2012 |
|
RU2495132C1 |
| Способ и набор для генодиагностики коклюша и коклюшеподобных заболеваний | 2018 |
|
RU2702240C1 |
| СПОСОБ И НАБОР ДЛЯ УСКОРЕННОЙ ЛАБОРАТОРНОЙ ДИАГНОСТИКИ КОКЛЮШНОЙ ИНФЕКЦИИ | 2013 |
|
RU2542396C1 |
| СПОСОБ ДИФФЕРЕНЦИАЦИИ ШТАММОВ BORDETELLA PERTUSSIS | 2005 |
|
RU2299908C1 |
| Способ лиофильного высушивания аттенуированных бактерий B. pertussis, аттенуированная бактерия B. pertussis, штамм аттенуированных бактерий B. Pertussis, вакцина, лиофилизированный вакцинный препарат | 2017 |
|
RU2709657C2 |
| СПОСОБ ДИФФЕРЕНЦИРОВАНИЯ ШТАММОВ Corynebacterium diphtheriae биовара gravis | 2007 |
|
RU2351656C1 |
| Свежевыделенный штамм бактерий Bordetella pertussis - продуцент комплекса протективных антигенов для производства бесклеточной коклюшной вакцины | 2018 |
|
RU2689903C1 |
| СПОСОБ ГЕНОТИПИРОВАНИЯ ИЗОЛЯТОВ NEISSERIA GONORRHOEAE НА БИОЛОГИЧЕСКОМ МИКРОЧИПЕ | 2023 |
|
RU2816767C1 |
| СПОСОБ ОПРЕДЕЛЕНИЯ СУБЛИНИЙ ВОЗБУДИТЕЛЯ ТУБЕРКУЛЕЗА ЛИНИИ L2 Beijing НА БИОЛОГИЧЕСКИХ МИКРОЧИПАХ | 2022 |
|
RU2790296C1 |
| НАБОР ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ ДЛЯ ГЕНОТИПИРОВАНИЯ ГРИБОВ ВИДОВОГО КОМПЛЕКСА COLLETOTRICHUM ACUTATUM | 2022 |
|
RU2807352C1 |
Изобретение относится к области микробиологии. Описан способ генотипирования В. pertussis из клинических образцов, отличающийся тем, что последовательно выполняют взятие биологического материала из ротоглотки больного двумя тампонами с последующим помещением их в физиологический раствор. Следом идёт выделение ДНК, проведение вложенной ПЦР с описанными праймерами оценка результатов путем электрофореза продуктов ПЦР в горизонтальном агарозном геле и проведения мультилокусного антигенного сиквенс-типирования по схеме MAST-2 для выявления аллельных вариантов фрагментов генов ptxP-fim3-prn, кодирующих промоторную ptxP область коклюшного токсина, фимбриальный Fim3 белок и пертактин. Технический результат заключается в разработке способа генотипирования B. pertussis по схеме MAST2 непосредственно из клинических образцов без выделения чистой культуры возбудителя на основе nested-ПЦР. 1 ил., 1 табл., 1 пр.
Способ генотипирования В. pertussis из клинических образцов, отличающийся тем, что последовательно выполняют следующие этапы:
- взятие биологического материала из ротоглотки больного двумя тампонами с последующим помещением их в физиологический раствор;
- выделение ДНК;
- проведение вложенной ПЦР с праймерами PtxP1wide ATATCCCGAATTCGTCGCCT, PtxP2wide GAGCACATTGTCGTTGTTTCC, PtxP1 AATCGTCCTGCTCAACCGCC, PtxP2 GGTATACGGTGGCGGGAGGA, Fim3Fwide CATGCTAATGGCCTCCGGT, Fim3Rwide GCGTGCTGAAGGACAAGATGG, Fim3F GACCTGATATTCTGATGCCG, Fim3R CGCAAGGCTTGCCGGTTTTTTTTGG, PrnFwide GGCGGTGTCTGTGTTGGG, PrnRwide GGTCAGCGTCAGCTTCAC, PrnC GTGCGGTTCCCGGCGGTG, PrnD GCTCCACGCTGGAGCCCG, PrnE TGCGGTTCCCGGCGGCTTC;
- последующая оценка результатов путем электрофореза продуктов ПЦР в горизонтальном агарозном геле и проведения мультилокусного антигенного сиквенс-типирования по схеме MAST-2 для выявления аллельных вариантов фрагментов генов ptxP-fim3-prn, кодирующих промоторную ptxP область коклюшного токсина, фимбриальный Fim3 белок и пертактин.
| СПОСОБ МОЛЕКУЛЯРНО-ГЕНЕТИЧЕСКОГО ТИПИРОВАНИЯ ШТАММОВ Bordetella pertussis | 2012 |
|
RU2495132C1 |
| СПОСОБ ДИФФЕРЕНЦИАЦИИ ШТАММОВ BORDETELLA PERTUSSIS | 2005 |
|
RU2299908C1 |
| Borisova O et al | |||
| Antigenic divergence between Bordetella pertussis clinical isolates from Moscow, Russia, and vaccine strains | |||
| Clin Vaccine Immunol | |||
| Пресс для выдавливания из деревянных дисков заготовок для ниточных катушек | 1923 |
|
SU2007A1 |
| Muyldermans G et al | |||
| Simple Algorithm for Identification of Bordetella pertussis Pertactin Gene Variants | |||
| J Clin | |||
Авторы
Даты
2024-07-04—Публикация
2023-10-05—Подача