Область техники
Настоящее изобретение относится к области биохимии, биотехнологии, молекулярной биологии, фитопатологии, в частности относится к контролю распространения возбудителя антракноза Colletotrichum lupini. В частности, относится к селекции люпина белого и люпина желтого на устойчивость к антракнозу, где используется для контроля инфекционного фона.
Уровень техники
Антракноз люпина, вызываемый по всему миру несовершенными грибами вида Colletotrichum lupini, обладающими высокой агрессивностью и вирулентностью по отношению растению-хозяину и приводящий к потерям до 90% урожая в отдельные годы, остается неразрешенной проблемой (Thomas G., et al., Application of fungicides to reduce yield loss in anthracnose-infected lupins. Crop Prot. 2008; 27, 1071–1077). Это препятствует включению в севооборот и увеличению посевных площадей люпина белого и люпина желтого. На сегодняшний момент проводится активный мониторинг распространения возбудителя антракноза по районам возделывания люпина, с целью определения регионов видообразования Colletotrichum lupini и определения карантинных зон, из-за наличия в них наиболее вредоносных изолятов гриба. Одной из основных задач для селекционеров, занимающихся люпином, остается поиск источников устойчивости к антракнозу, а также определение локусов в геноме, связанных с устойчивостью к данному заболеванию (Alkemade J.A., et al., Genetic diversity of Colletotrichum lupini and its virulence on white and Andean lupin. Sci Rep 11, 13547 (2021). https://doi.org/10.1038/s41598-021-92953-y). Однако, чтобы ставить контролируемые эксперименты по картированию локусов устойчивости и контролировать инфекционный фон в селекционных питомниках, необходимо обладать инструментом для точного определения генотипов грибов вида C. lupini.
Род Colletotrichum (тип Ascomycota, подтип Sordariomycetes, порядок Glomerellales) содержит по меньшей мере 150 видов, разделенных на десять основных клад. Одним из крупнейших из них является видовой комплекс Colletotrichum acutatum, в который входит вид Colletotrichum lupini, включает грибковые патогены, поражающие большое разнообразие растений в естественных и управляемых экосистемах. Видовой комплекс имеет очень широкий спектр хозяев, и штаммы были связаны с болезнями более чем 90 родов растений и по меньшей мере трех видов насекомых (Lardner, R., et al., Morphological and Molecular Analysis of Colletotrichum Acutatum Sensu Lato. Mycological Research 103, no. 3 (March 1999): 275–85. https://doi.org/10.1017/S0953756298007023). Согласно опубликованным данным, для генотипирования изолятов антракноза используют тест-системы, основанные на классической ПЦР, ПЦР в реальном времени с гибридизационными пробами (TaqMan), секвенирование по Сэнгеру (Kamber, Tim, et al., A QPCR Assay for the Fast Detection and Quantification of Colletotrichum Lupini. Plants 10, no. 8 (July 28, 2021): 1548. https://doi.org/10.3390/plants10081548). Классические ПЦР системы основаны на отсутствии/наличии фрагмента ДНК заведомо известного размера, определяемого с использованием агарозного или акриламидного гель-электрофореза после проведения реакции. При наличии фрагмента требуемого размера диагностируется принадлежность изучаемого образца к виду Colletotrichum lupini. Также для генотипирования и решения филогенетичских задач были созданы системы, основанные на определении количества повторов в микросателлитных последовательностях (Pecchia, Susanna, et al., Molecular Detection of the Seed-Borne Pathogen Colletotrichum Lupini Targeting the Hyper-Variable IGS Region of the Ribosomal Cluster. Plants 8, no. 7 (July 14, 2019): 222. https://doi.org/10.3390/plants8070222). Для выявления абсолютного и относительного уровня инфицирования растения-хозяина патогеном используются ПЦР-системы в реальном времени, основанные на гибридизации пробы (TaqMan) с флюорофором и гасителем к комплементарной матрице. Для решения филогенетических задач наиболее точным способом генотипирования, однако имеющим свои ограничения, остается метод секвенирования по Сенгеру нескольких локусов (ITS, GAPDH, TUB2, CHS-1, ACT, HIS3, HMG, APN/MAT1) как совместно, так и отдельно и обладающих высоким полиморфизмом среди видового комплекса Colletotrichum acutatum (Dubrulle, Guillaume, et al., Phylogenetic Diversity and Effect of Temperature on Pathogenicity of Colletotrichum Lupini. Plant Disease 104, no. 3 (March 2020): 938–50. https://doi.org/10.1094/PDIS-02-19-0273-RE). Однако в случае необходимости генотипирования большого количества образцов затраты на секвенирование методом Сенгера всех перечисленных локусов будут существенными. Так же данный метод возможно использовать только на моноспоровых культурах или линиях грибов, из-за этого время, требующееся для проведения анализа, значительно увеличивается.
Сущность изобретения
Одним из способов решения вышеозначенной проблемы является разработка набора олигонуклеотидных праймеров, специфически комплементарных консервативным участкам геномов грибов видового комплекса Colletotrichum acutatum, подходящих для идентификации сельскохозяйственно значимых видов рода Colletotrichum, в том числе возбудителя антракноза люпина белого и люпина желтого Colletotrichum lupini, продукты амплификации которых обладают размером не более 400 п. н., и их возможно использовать для последующего секвенирования нового поколения.
Задача определения видовой принадлежности грибов видового комплекса Colletotrichum acutatum решается путем создания набора специфичных праймеров, представляющими собой набор из олигонуклеотидных последовательностей длинной от 20 до 30 дезокирибонуклеотидов, к консервативным участкам локусов, обладающих видоспецифичными полиморфизмами и подходящими для секвенирования нового поколения (GTseq, AmpSeq, CleanPlex и др.).
Описанный в данном изобретении набор олигонуклеотидов разработан на основании анализа нуклеотидного разнообразия вариабельных участков моноспоровых линий грибов, в том числе патогенных, видового комплекса Colletotrichum acutatum.
Указанная задача решается путем создания набора праймеров для определения видовой принадлежности изолятов и/или штаммов грибов видового комплекса Colletotrichum acutatum, включающего следующие стадии:
1. Выделяют общую ДНК из образца или образцов, предположительно содержащих мицелий гриба.
2. К патентуемым праймерам (SEQ _ID_NO:1, SEQ _ID_NO:2, SEQ _ID_NO:3, SEQ _ID_NO:4, SEQ _ID_NO:5, SEQ _ID_NO:6, SEQ _ID_NO:7, SEQ _ID_NO:8, SEQ _ID_NO:9, SEQ _ID_NO:10, SEQ _ID_NO:11, SEQ _ID_NO:12, SEQ _ID_NO:13, SEQ _ID_NO:14, SEQ _ID_NO:15, SEQ _ID_NO:16, SEQ _ID_NO:17, SEQ _ID_NO:18, SEQ _ID_NO:19, SEQ _ID_NO:20) со стороны 5’- конца добавляется служебная последовательность, необходимая для присоединения технических последовательностей (последовательностей-адаптеров) (Yang, S., Fresnedo-Ramírez, J., Wang, M. et al. A next-generation marker genotyping platform (AmpSeq) in heterozygous crops: a case study for marker-assisted selection in grapevine. Hortic Res 3, 16002 (2016). https://doi.org/10.1038/hortres.2016.2; Campbell, N.R., Harmon, S.A. and Narum, S.R. (2015), Genotyping-in-Thousands by sequencing (GT-seq): A cost effective SNP genotyping method based on custom amplicon sequencing. Mol Ecol Resour, 15: 855-867. https://doi.org/10.1111/1755-0998.12357), зависящих от типа приготовляемой ДНК-библиотеки для секвенирования нового поколения и для последующего демультиплексирования и анализа образцов.
3. Методом мультиплексной ПЦР с использованием набора праймеров создается ДНК-библиотека ампликонов для всех исследуемых образцов.
4. Методом секвенирования нового поколения определяется последовательность нуклеотидов для каждого ампликона с двух сторон с длинной прочтений с каждой стороны не менее 150 нуклеотидов одновременно для всех образцов.
5. Биоинформатическими методами выравнивания последовательностей и филогенетического сравнения c генетическими базами данных, содержащими последовательности по локусам ITS, GAPDH, TUB2, APN/MAT1 для грибов видового комплекса Colletotrichum acutatum, определяется качественный видовой и внутривидовой состав в анализируемых образцах.
В некоторых вариантах изобретения данный способ характеризуется тем, что для каждого из образцов последовательности локусов ITS, GAPDH, TUB2, APN/MAT1 или каждого из локусов в отдельности можно определить с помощью секвенирования по Сэнгеру, но без необходимости добавления к 5’-концам праймеров технических последовательностей.
Таким образом, в рамках настоящего изобретения разработан набор олигонуклеотидных последовательностей, праймеров, для идентификации сельскохозяйственно значимых изолятов гриба Colletotrichum lupini, являющегося возбудителем антракноза люпина белого и люпина желтого, методом секвенирования ДНК путем амплификации и последующего определения нуклеотидных последовательностей фрагментов внутреннего транскрибируемого региона и 5.8S рРНК между генами малой (18S), большой субъединицы (23S) рибосомальной РНК (ITS1, ITS2), гена глицеральдегид-3-фосфатдегидрогеназа (GAPDH), гена бета-тубулина (TUB2), межгенной последовательности Apn2-Mat1-2-1 (APN/MAT1). Указанный набор содержит 20 олигонуклеотидов, обладающих праймерной активностью, комплементарных нуклеотидным последовательностям консервативных участков перечисленных локусов (ITS, GAPDH, TUB2, APN/MAT1) грибов видового комплекса Colletotrichum acutatum.
Настоящее изобретение позволяет расширить набор средств для амплификации и прицельного секвенирования, в том числе с применением методов секвенирования нового поколения фрагментов локусов (ITS, GAPDH, TUB2, APN/MAT1) различных изолятов вида Colletotrichum lupini, возбудителя антракноза люпина белого и люпина желтого, а также близких ему видов комплекса Colletotrichum acutatum (C.tamarilloi, C.costaricensis, C.melonis, C.cuscutae, C.paranaense, C.nymphaeae, C.simmondsii, C.acutatum).
Также изобретение позволяет повысить точность и скорость установления видовой принадлежности моноспоровых и мультиспоровых изолятов видов комплекса Colletotrichum acutatum, изучения их филогенетического родства на видовом уровне и идентификации сельскохозяйственно значимых изолятов антракноза, фитосанитарного контроля распространения патогенных штаммов, контроля инфекционного фона на селекционных и семеноводческих участках люпина белого и люпина желтого.
Краткое описание рисунков
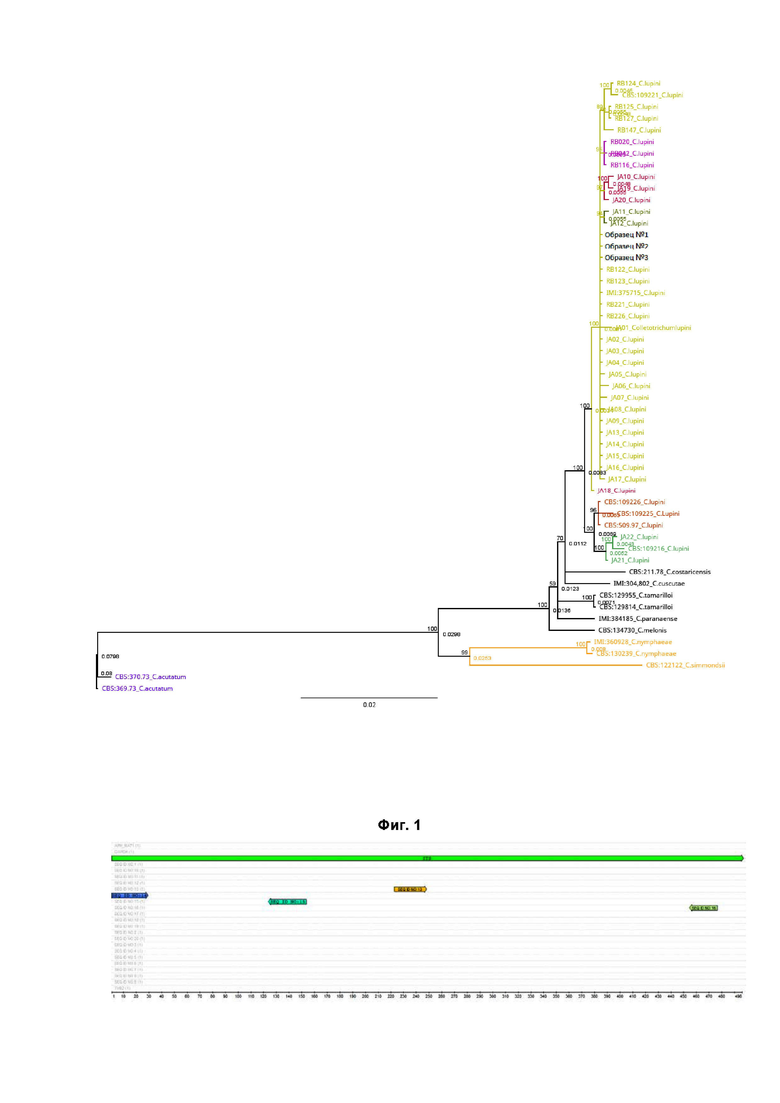
Фигура 1. Филогенетическое дерево, с исследуемыми образцами и образцами сравнения из видового комплекса Colletotrichum acutatum, построенное на основании нуклеотидных последовательностей локусов ITS, GAPDH, TUB2, APN/MAT1
Фигура 2. Схема расположения набора прямых и обратных праймеров на локусах а) ITS б) GAPDH в) TUB2 г) APN/MAT1
Подробное раскрытие изобретения
В описании данного изобретения термины «включает» и «включающий» интерпретируются как означающие «включает, помимо всего прочего». Указанные термины не предназначены для того, чтобы их истолковывали как «состоит только из». Если не определено отдельно, технические и научные термины в данной заявке имеют стандартные значения, общепринятые в научной и технической литературе.
Изолятом гриба называется первое односпоровое (моноспоровое) или чистое выделение грибов из любого источника, субстрата. Линия - внешне однородная популяция, выравненность которой поддерживается отбором. Термин «линия» применяют к группе особей, отличающихся от остальных характерным признаком или группой признаков и сохраняющих это отличие в течение ряда поколений. Обычно большинство особей линии гомозиготно по генам, определяющим характерные для нее признаки, но не исключено наличие гетерозигот по другим признакам. Штамм - чистая культура бактерий, грибов и иных микроорганизмов, выделенная из определенного источника и идентифицированная по тестам современной классификации. В рамках описания данного изобретения, термины изолят, линия и штамм являются взаимозаменяемыми, поскольку раскрываемый в данном изобретении способ определения вида на основе подобранного набора праймеров применим как к различным изолятам, так и к линиям и штаммам грибов видового комплекса Colletotrichum acutatum.
Номера нуклеотидных последовательностей (SEQ ID NO) в тексте описания соответствуют таковым в Перечне последовательностей согласно стандарту ST.26, являющемуся частью настоящего описания изобретения. В случае наличия разночтений в структуре последовательностей между текстом описания и соответствующей последовательностью в Перечне последовательностей согласно стандарту ST.26, приоритетными являются данные, представленные в тексте описания.
Данное изобретение описывает набор праймеров для установления видовой принадлежности изолятов грибов рода видового комплекса Colletotrichum acutatum.
Описанный в данном изобретении набор праймеров создан на основании анализа естественной вариабельности в популяции штаммов грибов рода видового комплекса Colletotrichum acutatum, выделенных с различных растений-хозяев в различных регионах Мира. Позиции под праймеры были выбраны в консервативных участках локусов ITS, GAPDH, TUB2, APN/MAT1, представленных в (Фигура 2), и являются специфичными. Решение, предлагаемое в данном изобретении, состоит в создании набора праймеров, при помощи которых в процессе ПЦР образуются ампликоны длиной не более 400 нуклеотидов, пригодные для секвенирования нового поколения с длиной прочтения не менее 150 пар нуклеотидов при прочтении в обе стороны. Такой набор праймеров позволяет в относительно короткие сроки создавать ДНК-библиотеку из десятков образцов изолятов грибов видового комплекса Colletotrichum acutatum и секвенировать ее методом нового поколения.
Олигонуклеотидные последовательности были разработаны in silico на основании консервативной референсной последовательности, указанной в Таблице 2, полученной при выравнивании последовательностей локусов ITS, GAPDH, TUB2, APN/MAT1 программой UGENE V44.0 алгоритмом MUSCLE V5, взятых из базы нуклеотидных последовательностей GenBank, для 50 штаммов грибов рода Colletotrichum, перечисленных в Таблице 1. Также олигонуклеотидные последовательности отбирали с учетом гуанин-цитозинового состава – от 40 до 60 %. Температуры плавления подбираемых праймеров находились в пределах от 55°C до 60°C. Для определения последовательностей ампликонов используют метод AmpSeq или подобный ему, включающем два раунда ПЦР. В первом раунде происходит амплификация вариабельного участка и добавление линкерных последовательностей, а во втором к линкерным последовательностям добавляют уникальные для каждого образца баркоды. Полученный пул ампликонов секвенируется по протоколу Illumina. Анализ результатов секвенирования проводится стандартными биоинформатическими методами. После чего последовательности от каждого образца выравниваются, собираются в одну и подвергаются филогенетическому анализу совместно с последовательностями 50 штаммов различных видов видового комплекса Colletotrichum acutatum (C. lupini, C. tamarilloi, C. costaricensis, C. melonis, C. cuscutae, C. paranaense, C. nymphaeae, C. simmondsii, C. acutatum) или аналогичным набором последовательностей. На основании проведенного выравнивания были рассчитаны апостериорные вероятности по алгоритму MrBayes с применением цепей Маркова и построено филогенетическое дерево в программном обеспечении UGENE V44.0. В полученной филограмме определяется в какую кладу, то есть к каким видами, попадет исследуемый образец, и на этом основании определяется его вид.
Структура 20 олигонуклеотидных последовательностей (10 пар праймеров в сочетаниях SEQ ID NO:14-SEQ ID NO:15; SEQ ID NO:13-SEQ ID NO:16; SEQ ID NO:11-SEQ ID NO:12; SEQ ID NO:17-SEQ ID NO:19; SEQ ID NO:18-SEQ ID NO:20; SEQ ID NO:2-SEQ ID NO:7; SEQ ID NO:6-SEQ ID NO:8; SEQ ID NO:1-SEQ ID NO:9; SEQ ID NO:3-SEQ ID NO:5; SEQ ID NO:4-SEQ ID NO:10):
SEQ ID NO:1
CGGTACTTTTATATGGAGCAGTGATTGG
SEQ ID NO:2
GCCGGGCGTCAACTGTGGTAAGTCT
SEQ ID NO:3
ATTGCTCTATCCTTTCGAGTCTTGCTCT
SEQ ID NO:4
TAAGTTAGCAGAAGAAGTCTCTTTTGCT
SEQ ID NO:5
TTCGTCTGCTGCCTTAGGCCGA
SEQ ID NO:6
GACCTGCCTATGTGGATCATCAGCTAT
SEQ ID NO:7
GAAATGTATAGACTACGACGCTTGA
SEQ ID NO:8
ATAATGGAATCATAATGTTCATGTCTCATT
SEQ ID NO:9
CGAAAGGATAGAGCAATTCGCGT
SEQ ID NO:10
AACCGGAGGAGCACTTCAGCCT
SEQ ID NO:11
TTCATTGAGACCAAGTACGCTGT
SEQ ID NO:12
GAGCGTACTTGAGCATGTAGGCCT
SEQ ID NO:13
GAACGCAGCGAAATGCGATAAGTAA
SEQ ID NO:14
TGAGTTACCGCTCTATAACCCTTTGTGA
SEQ ID NO:15
GACGTCGTGTAAATAGAGTTTGGTTTCCT
SEQ ID NO:16
TTTACGGCAAGAGTCCCTCCG
SEQ ID NO:17
GGTAACCAGATTGGTGCTGCCTTCT
SEQ ID NO:18
GGTCTCGACAGCAATGGCGTGTATG
SEQ ID NO:19
TAGACGCTCATGCGCTCGAGCTG
SEQ ID NO:20
GGAACGTACTTGTTGCCGGAGGCCT
Также каждому праймеру было присвоено название:
SEQ ID NO:1 - LCG_Sk_F1; SEQ ID NO:2 - LCG_Sk_F10; SEQ ID NO:3 - LCG_Sk_F11; SEQ ID NO:4 - LCG_Sk_F12; SEQ ID NO:5 - LCG_Sk_R10; SEQ ID NO:6 - LCG_Sk_R3; SEQ ID NO:7 - LCG_Sk_R4; SEQ ID NO:8 - LCG_Sk_R5; SEQ ID NO:9 - LCG_Sk_R6; SEQ ID NO:10 - LCG_Sk_R7; SEQ ID NO:11 - LCG_Sk_F5; SEQ ID NO:12 - LCG_Sk_R2; SEQ ID NO:13 - LCG_Sk_F2; SEQ ID NO:14 - LCG_Sk_F9; SEQ ID NO:15 - LCG_Sk_R1; SEQ ID NO:16 - LCG_Sk_R8; SEQ ID NO:17 - LCG_Sk_F6; SEQ ID NO:18 - LCG_Sk_F7; SEQ ID NO:19 - LCG_Sk_R11; SEQ ID NO:20 - LCG_Sk_R9.
(SEQ ID NO:21)
(SEQ ID NO:22)
(SEQ ID NO:23)
(SEQ ID NO:24)
Нижеследующие примеры осуществления способа приведены в целях раскрытия характеристик настоящего изобретения и их не следует рассматривать как каким-либо образом ограничивающие объем изобретения.
Пример 1. Применение набора праймеров для определения вида изолятов грибов видового комплекса Colletotrichum acutatum методом секвенирования нового поколения
Для демонстрации возможности использования набора праймеров при определении вида изолятов грибов видового комплекса Colletotrichum acutatum, были взяты 3 образца мицелия реизолированного с листьев, семян и створок бобов, с признаками антракноза, на картофельно-декстрозном агаре и морфологически подходящих под описание грибов рода Colletotrichum. С использованием набора праймеров были определены нуклеотидные последовательности локусов ITS, GAPDH, TUB2, APN/MAT1 у трех образцов сои на платформе MiniSeq Illumina. Этапы определения вида описаны ниже.
1. На первом этапе были выделены препараты ДНК из воздушного мицелия трех образцов, реизолированных со створок бобов и семян на картофельно-декстрозном агаре, собранных в полях люпина белого.
2. На втором этапе была проведена подготовка образцов ДНК гриба к дальнейшей работе, которая включала несколько последовательных этапов:
a) Мультиплексная ПЦР с набором из 10 пар праймеров для каждого образца в отдельности. При прохождении реакции происходит многократное увеличение количества вариабельных участков локусов ITS, GAPDH, TUB2, APN/MAT1. Этапы и условия проведения реакции включали: первичную денатурацию один цикл 1 минуту; 35 циклов состоящих из денатурации 30 секунд при 95°C, отжига праймеров при 58°C 30 секунд, элонгация при 72°C 30 секунд; 1 цикл 5 минут элонгации при 72°C.
b) Проведение второй реакции ПЦР для добавления индексных последовательностей к ампликонам каждого образца в отдельности. В качестве праймеров используются известные, специально подобранные, последовательности, которые совпадают с последовательностями на 5'-концах ампликонов, получившихся в результате первой ПЦР-реакции. Целью данного этапа является добавление индивидуальных последовательностей (индексов) к каждому ампликону, так как в последующем, при одновременном секвенировании всех образцов, остается возможность отнесения полученных последовательностей ДНК к определенному образцу изолята гриба.
c) Очистка каждого из 3 пулов ампликонов от ненужных на последующих этапах солей, неиспользованных нуклеотидов и т.д., с применением силико-колонок (Qiagen) и их перерастворение в 0,1х TE буфере.
d) Для получения библиотеки выполнили равномерное (по концентрации) смешивание трех пулов ампликонов.
3. В последующем, библиотека подготавливалась и секвенировалась согласно протоколам производителя секвенатора, в данном случае - Illumina.
Результаты определения последовательностей, полученных с помощью набора праймеров для локусов ITS, GAPDH, TUB2, APN/MAT1, трех образцов в соединенном виде приведены в Таблице 3.
образца
(SEQ ID NO:25)
(SEQ ID NO:26)
(SEQ ID NO:27)
В последующем последовательности образцов были выравнены с использованием алгоритма MUSCLE V5 совместно с 50 другими последовательностями из Таблицы 1, относящимися к видам C. lupini, C. tamarilloi, C. costaricensis, C. melonis, C. cuscutae, C. paranaense, C. nymphaeae, C. simmondsii, C. acutatum. На основании проведенного выравнивания были рассчитаны апостериорные вероятности по алгоритму MrBayes с применением цепей Маркова и построено филогенетическое дерево в программном обеспечении UGENE V44.0.
Последовательности образцов №1, №2 и №3 попали в кладу, содержащую последовательности, принадлежащие к разным по происхождению моноспоровым линиям гриба Colletotrichum lupini (Фиг. 2). Это позволило нам определить вид грибов в искомых образцах как Colletotrichum lupini. Полученные последовательности исследуемых образцов не содержали внутри образца вариабельности и были одинаковыми между собой, из чего можно сделать вывод о том, что собранные изоляты одинаковы или очень близки по генотипу. Полученные результаты показывают возможность успешного применения представленного набора праймеров для определения видовой принадлежности изолятов в видовом комплексе Colletotrichum acutatum методом секвенирования нового поколения.
Пример 2. Применение набора праймеров для определения вида изолятов грибов видового комплекса Colletotrichum acutatum методом севенирования по Сэнгеру
Для демонстрации возможности использования набора праймеров для определения последовательности нуклеотидов моноспоровой культуры гриба видового комплекса Colletotrichum acutatum, был взят 1 образец мицелия реизолированного с семян с признаками антракноза, на картофельно-декстрозном агаре и морфологически подходящего под описание. С использованием набора праймеров для определения вида грибов были определены нуклеотидные последовательности локусов ITS, GAPDH, TUB2, APN/MAT1 у образца методом секвенирования по Сэнгеру. Этапы определения последовательности описаны ниже:
1. На первом этапе был выделен препарат ДНК из воздушного мицелия моноспоровой культуры, реизолированной с зараженных антракнозом семян люпина белого.
2. На втором этапе была проведена подготовка образца ДНК гриба к дальнейшей работе, которая включала несколько последовательных этапов:
a) Провели ПЦР с каждой из 10 пар праймеров в отдельной пробирке с ДНК интересующего образца. При прохождении реакции происходит многократное увеличение количества вариабельных участков локусов ITS, GAPDH, TUB2, APN/MAT1. Этапы и условия проведения реакции включали первичную денатурацию один цикл 1 минуту; 35 циклов состоящих из денатурации 30 секунд при 95°C, отжига праймеров при 58°C 30 секунд, элонгация при 72°C 30 секунд; 1 цикл 5 минут элонгации при 72°C.
b) Очистили продукты всех ПЦР, проведенных в предыдущем шаге, от ненужных на последующих этапах солей, неиспользованных нуклеотидов и т.д., с применением силико-колонок (Qiagen), и их перерастворение в 0,1х TE буфере.
c) Провели вторую реакцию терминирующего секвенирования для всех продуктов ПЦР с соответствующими им праймерами.
3. Провели отдельный капиллярный электрофорез для всех продуктов реакций терминирующего секвенирования.
Собранная последовательность локусов ITS, GAPDH, TUB2, APN/MAT1, для образца в объединенном виде приведена в Таблице 4.
Образца
(SEQ ID NO:28)
В последующем последовательности образцов были выравнены с использованием алгоритма MUSCLE V5 совместно с 50 другими последовательностями из Таблицы 1, относящимися к видам C. lupini, C. tamarilloi, C. costaricensis, C. melonis, C. cuscutae, C. paranaense, C. nymphaeae, C. simmondsii, C. acutatum. На основании проведенного выравнивания были рассчитаны апостериорные вероятности по алгоритму MrBayes с применением цепей Маркова и построено филогенетическое дерево в программном обеспечении UGENE V44.0.
Последовательности образца №1 попали в кладу, содержащую последовательности принадлежащие к разным по происхождению моноспоровым линиям гриба Colletotrichum lupini. Это позволило нам определить вид грибов в искомых образцах как Colletotrichum lupini.
Полученные результаты показывают возможность успешного применения представленного набора праймеров для амплификации и последующего определения последовательности ДНК в локусах ITS, GAPDH, TUB2, APN/MAT1 моноспоровых культур видового комплекса Colletotrichum acutatum.
--->
This XML file does not appear to have any style information
associated with it. The document tree is shown below.
<ST26SequenceListing dtdVersion="V1_3" fileName="Colletotrichum
acutatum.xml" softwareName="WIPO Sequence" softwareVersion="2.0.0"
productionDate="2022-12-16">
<ApplicantFileReference>481972</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное научное учреждение "Всероссийский научно-исследовательский
институт радиологии и агроэкологии"</ApplicantName>
<ApplicantNameLatin>Federal State Budgetary Scientific Institution
"All-Russian Research Institute of Radiology and
Agroecology"</ApplicantNameLatin>
<InventionTitle languageCode="ru">Набор олигонуклеотидных праймеров
для генотипирования грибов видового комплекса Colletotrichum
acutatum</InventionTitle>
<SequenceTotalQuantity>28</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>28</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..28</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cggtacttttatatggagcagtgattgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gccgggcgtcaactgtggtaagtct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>28</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..28</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q3">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>attgctctatcctttcgagtcttgctct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>28</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..28</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>taagttagcagaagaagtctcttttgct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttcgtctgctgccttaggccga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gacctgcctatgtggatcatcagctat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q7">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaaatgtatagactacgacgcttga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ataatggaatcataatgttcatgtctcatt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q9">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgaaaggatagagcaattcgcgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaccggaggagcacttcagcct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q11">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttcattgagaccaagtacgctgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagcgtacttgagcatgtaggcct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q13">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaacgcagcgaaatgcgataagtaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>28</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..28</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgagttaccgctctataaccctttgtga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>29</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q15">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gacgtcgtgtaaatagagtttggtttcct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tttacggcaagagtccctccg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q17">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtaaccagattggtgctgccttct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtctcgacagcaatggcgtgtatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q19">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tagacgctcatgcgctcgagctg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggaacgtacttgttgccggaggcct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>496</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..496</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgagttaccgctctataaccctttgtgaacntacctaaccgttgcttcggcg
ggcaggggaagcctctcgcgggccntcccctcccggcgccgnnccccaccacggggacggggcgcccgcc
ggaggaaaccaaactctatttacacgacgtctcttctgagtggcacaagcaaataattaaaacttttaac
aacggatctcttggttctggcatcgatgaagaacgcagcgaaatgcgataagtaatgngaattgcagaat
tcagtgaatcatcgaatctttgaacgcacattgcgctcgccagcattctggcgagcatgcctgttcgagc
gtcatttcaaccctcaagcaccgcttggttttggggccccacggcanacgtgggcccttnaaggtagtgg
cggaccctcccggagcctcctttgcgtagtaactaacgnctcgcacngggatncggagggactcttgccg
taaancccccnnanttntttacag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>259</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..259</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q23">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttcattgagaccaagtacgctgtnagtancanccnactttacccctccatcn
tgntatcncgtctnccacnataacaccagcttcgtcgntancnncnggnaaaagagtnngnnctagcnnn
ctcnacntntttnnccccnnggtttcgantgggctngttgtannganncgacgtganacaancntgcnga
aacanccnagacnnaanttgctgacnagacanatnnnncacnaggcctacatgctcaagtacgctcc</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="23">
<INSDSeq>
<INSDSeq_length>431</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..431</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtaaccagattggtgctgccttctggtgcgtagccaaccgccnncgangcg
gcgatnnnganatttgacacgatctcgnactnannttngnnacaggcanaacatctctggcgagcacggt
ctcgacagcaatggcgtgtatgtcncnngtcctcnagngnggcnnccncntggaccnannagctaatcan
nncanaggtacaacggcacttccgagctncagctcgagcgcatgagcgtctanttcaacgaagtttgtta
ncctagtnccccagtgtgcaggcaancctattgacgaatgctgaccttctcacccaaccaggcctccggc
aacaagtacgttccncgcgccgtnctcgtcgacttggagcccggtaccatggacgccgtccgtgccggtc
ccttnggccancttttccgccccgacaac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="24">
<INSDSeq>
<INSDSeq_length>1183</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1183</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q25">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gccgggcgtcaactgtggtaagtctanacnnnnnnnnnnnnnccaacnnnnn
nnnnnnncacncnnnncanacanacgnccnnanccccttnttnnnctcntttatgcgnccaccgaggccn
nnngnccnaccctcntctcnntcnntnccccgacatatnccganctttcnngnnnnnnnnnnnnnnnnnn
ncnncnttnatagctgatgatccacataggcaggtcnnttntncatntgcgcncgtcctntggggaaatc
nggagaaaaggaaaangngacggagtggagntgcggtacttttatatggagcagtgattggaanaanagc
cannntcaagcgtcgtagtctatacatttcnnaaggngcganntnnagncnngcgnnantcgnngnanng
nntgtnangagntgnnnncntnacnntngaaaanagananntnggtantttagnngtgcagaanaagang
atnnannnaatgagacatgaacattatgattccattatngttnnngnaantctntcaancantntatatc
gntnttgtgntngctcatcntnnnatcatggcagnnntagacatccnanncttnggcancnannacgcga
attgctctatcctttcgagtcttgctctnntngnagtngcncactntttgcntgaacnccatcaagagng
ncgcnaccgncnccncncnatnatncgtccnnnatnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnn
nnnnnnnnnnnnnnnnnnnnncncnaannnnagcctntcntngnctnnannggnccntaagttagcagaa
gaagtctcttttgctnnngctgannnnnnnnnnnnnnnnnnnnnnncacggcatgtctncttagtnntng
cccnancntcnngtgntggtgttntttngacttnctnggcgcnanctgggananggctntganggnagnn
gaggnanantcggcctaaggcagcagacgaaggtnctccttcgcngcaacttntantcgcnnannttntt
gcacngtnatngngacntngcanggtagnaacnnntgnagcgacgntgtgatggnncgnttcttgntccn
angatccttnnnctccatctnnanaacngngntgnnngngannntnaggctgaagtgctcctccggttgc
gacgacgcttc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="25">
<INSDSeq>
<INSDSeq_length>2313</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..2313</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgagttaccgctctataaccctttgtgaacgtacctaaccgttgcttcggcg
ggcaggggaagcctctcgcgggcctcccctcccggcgccggcccccaccacggggacggggcgcccgccg
gaggaaaccaaactctatttacacgacgtctcttctgagtggcacaagcaaataattaaaacttttaaca
acggatctcttggttctggcatcgatgaagaacgcagcgaaatgcgataagtaatgtgaattgcagaatt
cagtgaatcatcgaatctttgaacgcacattgcgctcgccagcattctggcgagcatgcctgttcgagcg
tcatttcaaccctcaagcaccgcttggttttggggccccacggcacacgtgggcccttgaaggtagtggc
ggaccctcccggagcctcctttgcgtagtaactaacgtctcgcactgggatccggagggactcttgccgt
aaaaccccccaattctttacagttcattgagaccaagtacgctgtgagtatcaccccactttacccctcc
atcatgatatcacgtctgccacgataacaccagcttcgtcggtacccacggcaaaagagtcagaactagc
accctcgacttttttgccccaaggtttcgattgggcttgttgtaatgacacgacgtgacacaatcatgcc
gaaacagccgagacaaaacttgctgacagacaatcatcacaggcctacatgctcaagtaggtaaccagat
tggtgctgccttctggtgcgtagccaaccgccaacgacgcggcgatttcgatatttgacacgatctcgaa
ctgaccttgatacaggcagaacatctctggcgagcacggtctcgacagcaatggcgtgtatgtcacttgt
cctccagtgcggcatcctcatggacccagcagctaatcacaccacaggtacaacggcacttccgagctcc
agctcgagcgcatgagcgtctatttcaacgaagtttgttatcctagtcccccagtgtgcaggcaatccta
ttgacgaatgctgaccttctcacccaaccaggcctccggcaacaagtacgttcctcgcgccgtcctcgtc
gacttggagcccggtaccatggacgccgtccgtgccggtccctttggccagcttttccgccccgacaacg
ccgggcgtcaactgtggtaagtctaaacccaaccacaccacacacacgccttacccccttcttactcgtt
tatgcgtccaccgaggcctctgtcctaccctcctctctctcgtcccccgacatatcccgatctttctgcc
cccgccccccctcgacccccattcatagctgatgatccacataggcaggtcgtttttcatctgcgcccgt
cctctggggaaatccggagaaaaggaaaaggggacggagtggaggtgcggtacttttatatggagcagtg
attggaaaaagagccagtctcaagcgtcgtagtctatacatttctaaggggcgatgtggagtcacgcgag
attcgacgaacagtgtgacgagctgtaaccgttactatcgaaaagagacagctaggtagtttagtcgtgc
agaagaagaagattaaggcaatgagacatgaacattatgattccattatcgttttagtaaatctatcaag
cattctatatcggtcttgtggtggctcatcttcgcatcatggcagagttagacatccgattcttcggcag
cgacgacgcgaattgctctatcctttcgagtcttgctctgcttgaagtagctcactctttgcttgaaccc
catcaagagcgtcgccaccgtctccgcccaatgatgcgtccggaattcgcccatcttgtgagtagagcag
gtaaaactccacgaagtggctttggagccttacacgaattcgagcctttcctggtctcgacaggccccta
agttagcagaagaagtctcttttgctccggctgagcatggtaagggtgagtgccgggcacggcatgtctg
cttagttctggcccaaacatcaagtgttggtgttttttcgactttctcggcgcgagctgggacatggctc
tgagggaagacgaggcatactcggcctaaggcagcagacgaaggtgctccttcgcggcaacttgtagtcg
cgaaaattcttgcacagtcattgagacctagcatggtaggaactaatgcagcgacgttgtgatggatcgc
ttcttgctcctacgatccttgatctccatctctagaacggggtggcagggacgattaggctgaagtgctc
ctccggttgcgacgacgcttc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="26">
<INSDSeq>
<INSDSeq_length>2313</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..2313</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q27">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgagttaccgctctataaccctttgtgaacgtacctaaccgttgcttcggcg
ggcaggggaagcctctcgcgggcctcccctcccggcgccggcccccaccacggggacggggcgcccgccg
gaggaaaccaaactctatttacacgacgtctcttctgagtggcacaagcaaataattaaaacttttaaca
acggatctcttggttctggcatcgatgaagaacgcagcgaaatgcgataagtaatgtgaattgcagaatt
cagtgaatcatcgaatctttgaacgcacattgcgctcgccagcattctggcgagcatgcctgttcgagcg
tcatttcaaccctcaagcaccgcttggttttggggccccacggcacacgtgggcccttgaaggtagtggc
ggaccctcccggagcctcctttgcgtagtaactaacgtctcgcactgggatccggagggactcttgccgt
aaaaccccccaattctttacagttcattgagaccaagtacgctgtgagtatcaccccactttacccctcc
atcatgatatcacgtctgccacgataacaccagcttcgtcggtacccacggcaaaagagtcagaactagc
accctcgacttttttgccccaaggtttcgattgggcttgttgtaatgacacgacgtgacacaatcatgcc
gaaacagccgagacaaaacttgctgacagacaatcatcacaggcctacatgctcaagtaggtaaccagat
tggtgctgccttctggtgcgtagccaaccgccaacgacgcggcgatttcgatatttgacacgatctcgaa
ctgaccttgatacaggcagaacatctctggcgagcacggtctcgacagcaatggcgtgtatgtcacttgt
cctccagtgcggcatcctcatggacccagcagctaatcacaccacaggtacaacggcacttccgagctcc
agctcgagcgcatgagcgtctatttcaacgaagtttgttatcctagtcccccagtgtgcaggcaatccta
ttgacgaatgctgaccttctcacccaaccaggcctccggcaacaagtacgttcctcgcgccgtcctcgtc
gacttggagcccggtaccatggacgccgtccgtgccggtccctttggccagcttttccgccccgacaacg
ccgggcgtcaactgtggtaagtctaaacccaaccacaccacacacacgccttacccccttcttactcgtt
tatgcgtccaccgaggcctctgtcctaccctcctctctctcgtcccccgacatatcccgatctttctgcc
cccgccccccctcgacccccattcatagctgatgatccacataggcaggtcgtttttcatctgcgcccgt
cctctggggaaatccggagaaaaggaaaaggggacggagtggaggtgcggtacttttatatggagcagtg
attggaaaaagagccagtctcaagcgtcgtagtctatacatttctaaggggcgatgtggagtcacgcgag
attcgacgaacagtgtgacgagctgtaaccgttactatcgaaaagagacagctaggtagtttagtcgtgc
agaagaagaagattaaggcaatgagacatgaacattatgattccattatcgttttagtaaatctatcaag
cattctatatcggtcttgtggtggctcatcttcgcatcatggcagagttagacatccgattcttcggcag
cgacgacgcgaattgctctatcctttcgagtcttgctctgcttgaagtagctcactctttgcttgaaccc
catcaagagcgtcgccaccgtctccgcccaatgatgcgtccggaattcgcccatcttgtgagtagagcag
gtaaaactccacgaagtggctttggagccttacacgaattcgagcctttcctggtctcgacaggccccta
agttagcagaagaagtctcttttgctccggctgagcatggtaagggtgagtgccgggcacggcatgtctg
cttagttctggcccaaacatcaagtgttggtgttttttcgactttctcggcgcgagctgggacatggctc
tgagggaagacgaggcatactcggcctaaggcagcagacgaaggtgctccttcgcggcaacttgtagtcg
cgaaaattcttgcacagtcattgagacctagcatggtaggaactaatgcagcgacgttgtgatggatcgc
ttcttgctcctacgatccttgatctccatctctagaacggggtggcagggacgattaggctgaagtgctc
ctccggttgcgacgacgcttc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="27">
<INSDSeq>
<INSDSeq_length>2313</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..2313</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgagttaccgctctataaccctttgtgaacgtacctaaccgttgcttcggcg
ggcaggggaagcctctcgcgggcctcccctcccggcgccggcccccaccacggggacggggcgcccgccg
gaggaaaccaaactctatttacacgacgtctcttctgagtggcacaagcaaataattaaaacttttaaca
acggatctcttggttctggcatcgatgaagaacgcagcgaaatgcgataagtaatgtgaattgcagaatt
cagtgaatcatcgaatctttgaacgcacattgcgctcgccagcattctggcgagcatgcctgttcgagcg
tcatttcaaccctcaagcaccgcttggttttggggccccacggcacacgtgggcccttgaaggtagtggc
ggaccctcccggagcctcctttgcgtagtaactaacgtctcgcactgggatccggagggactcttgccgt
aaaaccccccaattctttacagttcattgagaccaagtacgctgtgagtatcaccccactttacccctcc
atcatgatatcacgtctgccacgataacaccagcttcgtcggtacccacggcaaaagagtcagaactagc
accctcgacttttttgccccaaggtttcgattgggcttgttgtaatgacacgacgtgacacaatcatgcc
gaaacagccgagacaaaacttgctgacagacaatcatcacaggcctacatgctcaagtaggtaaccagat
tggtgctgccttctggtgcgtagccaaccgccaacgacgcggcgatttcgatatttgacacgatctcgaa
ctgaccttgatacaggcagaacatctctggcgagcacggtctcgacagcaatggcgtgtatgtcacttgt
cctccagtgcggcatcctcatggacccagcagctaatcacaccacaggtacaacggcacttccgagctcc
agctcgagcgcatgagcgtctatttcaacgaagtttgttatcctagtcccccagtgtgcaggcaatccta
ttgacgaatgctgaccttctcacccaaccaggcctccggcaacaagtacgttcctcgcgccgtcctcgtc
gacttggagcccggtaccatggacgccgtccgtgccggtccctttggccagcttttccgccccgacaacg
ccgggcgtcaactgtggtaagtctaaacccaaccacaccacacacacgccttacccccttcttactcgtt
tatgcgtccaccgaggcctctgtcctaccctcctctctctcgtcccccgacatatcccgatctttctgcc
cccgccccccctcgacccccattcatagctgatgatccacataggcaggtcgtttttcatctgcgcccgt
cctctggggaaatccggagaaaaggaaaaggggacggagtggaggtgcggtacttttatatggagcagtg
attggaaaaagagccagtctcaagcgtcgtagtctatacatttctaaggggcgatgtggagtcacgcgag
attcgacgaacagtgtgacgagctgtaaccgttactatcgaaaagagacagctaggtagtttagtcgtgc
agaagaagaagattaaggcaatgagacatgaacattatgattccattatcgttttagtaaatctatcaag
cattctatatcggtcttgtggtggctcatcttcgcatcatggcagagttagacatccgattcttcggcag
cgacgacgcgaattgctctatcctttcgagtcttgctctgcttgaagtagctcactctttgcttgaaccc
catcaagagcgtcgccaccgtctccgcccaatgatgcgtccggaattcgcccatcttgtgagtagagcag
gtaaaactccacgaagtggctttggagccttacacgaattcgagcctttcctggtctcgacaggccccta
agttagcagaagaagtctcttttgctccggctgagcatggtaagggtgagtgccgggcacggcatgtctg
cttagttctggcccaaacatcaagtgttggtgttttttcgactttctcggcgcgagctgggacatggctc
tgagggaagacgaggcatactcggcctaaggcagcagacgaaggtgctccttcgcggcaacttgtagtcg
cgaaaattcttgcacagtcattgagacctagcatggtaggaactaatgcagcgacgttgtgatggatcgc
ttcttgctcctacgatccttgatctccatctctagaacggggtggcagggacgattaggctgaagtgctc
ctccggttgcgacgacgcttc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="28">
<INSDSeq>
<INSDSeq_length>2313</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..2313</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q29">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgagttaccgctctataaccctttgtgaacgtacctaaccgttgcttcggcg
ggcaggggaagcctctcgcgggcctcccctcccggcgccggcccccaccacggggacggggcgcccgccg
gaggaaaccaaactctatttacacgacgtctcttctgagtggcacaagcaaataattaaaacttttaaca
acggatctcttggttctggcatcgatgaagaacgcagcgaaatgcgataagtaatgtgaattgcagaatt
cagtgaatcatcgaatctttgaacgcacattgcgctcgccagcattctggcgagcatgcctgttcgagcg
tcatttcaaccctcaagcaccgcttggttttggggccccacggcacacgtgggcccttgaaggtagtggc
ggaccctcccggagcctcctttgcgtagtaactaacgtctcgcactgggatccggagggactcttgccgt
aaaaccccccaattctttacagttcattgagaccaagtacgctgtgagtatcaccccactttacccctcc
atcatgatatcacgtctgccacgataacaccagcttcgtcggtacccacggcaaaagagtcagaactagc
accctcgacttttttgccccaaggtttcgattgggcttgttgtaatgacacgacgtgacacaatcatgcc
gaaacagccgagacaaaacttgctgacagacaatcatcacaggcctacatgctcaagtaggtaaccagat
tggtgctgccttctggtgcgtagccaaccgccaacgacgcggcgatttcgatatttgacacgatctcgaa
ctgaccttgatacaggcagaacatctctggcgagcacggtctcgacagcaatggcgtgtatgtcacttgt
cctccagtgcggcatcctcatggacccagcagctaatcacaccacaggtacaacggcacttccgagctcc
agctcgagcgcatgagcgtctatttcaacgaagtttgttatcctagtcccccagtgtgcaggcaatccta
ttgacgaatgctgaccttctcacccaaccaggcctccggcaacaagtacgttcctcgcgccgtcctcgtc
gacttggagcccggtaccatggacgccgtccgtgccggtccctttggccagcttttccgccccgacaacg
ccgggcgtcaactgtggtaagtctaaacccaaccacaccacacacacgccttacccccttcttactcgtt
tatgcgtccaccgaggcctctgtcctaccctcctctctctcgtcccccgacatatcccgatctttctgcc
cccgccccccctcgacccccattcatagctgatgatccacataggcaggtcgtttttcatctgcgcccgt
cctctggggaaatccggagaaaaggaaaaggggacggagtggaggtgcggtacttttatatggagcagtg
attggaaaaagagccagtctcaagcgtcgtagtctatacatttctaaggggcgatgtggagtcacgcgag
attcgacgaacagtgtgacgagctgtaaccgttactatcgaaaagagacagctaggtagtttagtcgtgc
agaagaagaagattaaggcaatgagacatgaacattatgattccattatcgttttagtaaatctatcaag
cattctatatcggtcttgtggtggctcatcttcgcatcatggcagagttagacatccgattcttcggcag
cgacgacgcgaattgctctatcctttcgagtcttgctctgcttgaagtagctcactctttgcttgaaccc
catcaagagcgtcgccaccgtctccgcccaatgatgcgtccggaattcgcccatcttgtgagtagagcag
gtaaaactccacgaagtggctttggagccttacacgaattcgagcctttcctggtctcgacaggccccta
agttagcagaagaagtctcttttgctccggctgagcatggtaagggtgagtgccgggcacggcatgtctg
cttagttctggcccaaacatcaagtgttggtgttttttcgactttctcggcgcgagctgggacatggctc
tgagggaagacgaggcatactcggcctaaggcagcagacgaaggtgctccttcgcggcaacttgtagtcg
cgaaaattcttgcacagtcattgagacctagcatggtaggaactaatgcagcgacgttgtgatggatcgc
ttcttgctcctacgatccttgatctccatctctagaacggggtggcagggacgattaggctgaagtgctc
ctccggttgcgacgacgcttc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ мультиплексной идентификации 32 генетических маркеров льна | 2022 |
|
RU2804939C1 |
| Способ мультиплексной идентификации 52 генетических маркеров хозяйственно ценных признаков льна | 2023 |
|
RU2826718C1 |
| Нуклеотидная последовательность, кодирующая фермент литиказу, и панель олигонуклеотидов для получения синтетической нуклеотидной последовательности гена литиказы | 2023 |
|
RU2826150C1 |
| Способ сполиготипирования микобактерий туберкулезного комплекса с использованием ДНК-амплификации в иммобилизованной фазе и биологический микрочип для его осуществления | 2023 |
|
RU2807998C1 |
| СПОСОБ ГЕНОТИПИРОВАНИЯ ИЗОЛЯТОВ NEISSERIA GONORRHOEAE НА БИОЛОГИЧЕСКОМ МИКРОЧИПЕ | 2023 |
|
RU2816767C1 |
| Набор праймеров для выявления возбудителей бактериальной пневмонии человека методом мультиплексной рекомбиназной полимеразной амплификации | 2023 |
|
RU2813995C1 |
| СПОСОБ ОПРЕДЕЛЕНИЯ СУБЛИНИЙ ВОЗБУДИТЕЛЯ ТУБЕРКУЛЕЗА ЛИНИИ L2 Beijing НА БИОЛОГИЧЕСКИХ МИКРОЧИПАХ | 2022 |
|
RU2790296C1 |
| Способ анализа соматических мутаций в генах IDH1 и IDH2 с использованием LNA-блокирующей ПЦР и гибридизации с биологическим микрочипом | 2024 |
|
RU2839291C1 |
| СПОСОБ ВЫДЕЛЕНИЯ ТОТАЛЬНОЙ ДНК БАКТЕРИЙ ИЗ ОБРАЗЦОВ ПОЧВЫ, СПОСОБ ОЦЕНКИ БАКТЕРИАЛЬНОГО СОСТАВА ПОЧВ ПОСРЕДСТВОМ МЕТАГЕНОМНОГО СЕКВЕНИРОВАНИЯ И НАБОРЫ ДЛЯ ОСУЩЕСТВЛЕНИЯ СПОСОБОВ | 2024 |
|
RU2829656C1 |
| Панель праймеров для полногеномного секвенирования вирусов парагриппа человека 1 и 2 типа | 2024 |
|
RU2833687C1 |

Настоящее изобретение относится к области биохимии, биотехнологии, молекулярной биологии. Предложен способ определения видовой принадлежности изолятов и/или штаммов грибов видового комплекса Colletotrichum acutatum в образце, включающий следующие этапы: выделяют общую ДНК из образца, предположительно содержащего мицелий гриба; проводят ПЦР с праймерами и методом секвенирования определяют последовательности нуклеотидов ампликонов локусов ITS, GAPDH, TUB2, APN/MAT1; методом выравнивания последовательностей и филогенетического сравнения c генетическими базами данных, содержащими последовательности по локусам ITS, GAPDH, TUB2, APN/MAT1 для грибов видового комплекса Colletotrichum acutatum, определяют качественный видовой и внутривидовой состав грибов рода Colletotrichum в анализируемом образце. Изобретение позволяет расширить набор средств для амплификации и прицельного секвенирования нового поколения фрагментов локусов (ITS, GAPDH, TUB2, APN/MAT1) различных изолятов вида Colletotrichum lupini, возбудителя антракноза люпина белого и люпина жёлтого, а также близких ему видов комплекса Colletotrichum acutatum (C.tamarilloi, C.costaricensis, C.melonis, C.cuscutae, C.paranaense, C.nymphaeae, C.simmondsii, C.acutatum). Также изобретение позволяет повысить точность и скорость установления видовой принадлежности моноспоровых и мультиспоровых изолятов видов комплекса Colletotrichum acutatum, изучения их филогенетического родства на видовом уровне и идентификации сельскохозяйственно значимых изолятов антракноза, фитосанитарного контроля распространения патогенных штаммов, контроля инфекционного фона на селекционных и семеноводческих участках люпина белого и люпина жёлтого. 3 з.п. ф-лы, 4 табл., 2 ил., 2 пр.
1. Способ определения видовой принадлежности изолятов и/или штаммов грибов видового комплекса Colletotrichum acutatum в образце, включающий следующие этапы:
a) выделяют общую ДНК из образца, предположительно содержащего мицелий гриба;
b) проводят ПЦР с праймерами SEQ ID NO:1, SEQ ID NO:2, SEQ ID NO:3, SEQ ID NO:4, SEQ ID NO:5, SEQ ID NO:6, SEQ ID NO:7, SEQ ID NO:8, SEQ ID NO:9, SEQ ID NO:10, SEQ ID NO:11, SEQ ID NO:12, SEQ ID NO:13, SEQ ID NO:14, SEQ ID NO:15, SEQ ID NO:16, SEQ ID NO:17, SEQ ID NO:18, SEQ ID NO:19 и SEQ ID NO:20 и методом секвенирования определяют последовательности нуклеотидов ампликонов локусов ITS, GAPDH, TUB2, APN/MAT1;
c) методом выравнивания последовательностей и филогенетического сравнения c генетическими базами данных, содержащими последовательности по локусам ITS, GAPDH, TUB2, APN/MAT1 для грибов видового комплекса Colletotrichum acutatum, определяют качественный видовой и внутривидовой состав грибов рода Colletotrichum в анализируемом образце.
2. Способ по п.1, характеризующийся тем, что на этапе b) последовательности нуклеотидов локусов ITS, GAPDH, TUB2, APN/MAT1 определяют с помощью секвенирования нового поколения, включающего следующие стадии:
1) к последовательности каждого праймера SEQ ID NO:1, SEQ ID NO:2, SEQ ID NO:3, SEQ ID NO:4, SEQ ID NO:5, SEQ ID NO:6, SEQ ID NO:7, SEQ ID NO:8, SEQ ID NO:9, SEQ ID NO:10, SEQ ID NO:11, SEQ ID NO:12, SEQ ID NO:13, SEQ ID NO:14, SEQ ID NO:15, SEQ ID NO:16, SEQ ID NO:17, SEQ ID NO:18, SEQ ID NO:19 и SEQ ID NO:20 со стороны 5’-конца добавляют служебную последовательность, обеспечивающую возможность присоединения последовательностей-адаптеров, зависящих от типа приготовляемой ДНК-библиотеки для секвенирования нового поколения и для последующего, при анализе, демультиплексирования образцов;
2) методом мультиплексной ПЦР с использованием набора праймеров создают ДНК-библиотеку ампликонов исследуемого образца;
3) методом секвенирования нового поколения определяют последовательность нуклеотидов для каждого ампликона с двух сторон с длиной прочтений с каждой стороны не менее 150 нуклеотидов одновременно для всех ампликонов.
3. Способ по п.1, характеризующийся тем, что на этапе b) последовательности нуклеотидов локусов ITS, GAPDH, TUB2, APN/MAT1 определяют при помощи секвенирования по Сэнгеру, включающего следующие стадии:
1) методом ПЦР проводят 10 отдельных реакций с парами праймеров SEQ ID NO:14-SEQ ID NO:15; SEQ ID NO:13-SEQ ID NO:16; SEQ ID NO:11-SEQ ID NO:12; SEQ ID NO:17-SEQ ID NO:19; SEQ ID NO:18-SEQ ID NO:20; SEQ ID NO:2-SEQ ID NO:7; SEQ ID NO:6-SEQ ID NO:8; SEQ ID NO:1-SEQ ID NO:9; SEQ ID NO:3-SEQ ID NO:5; SEQ ID NO:4-SEQ ID NO:10;
2) проводят секвенирование по Сэнгеру всех полученных продуктов ПЦР.
4. Способ по п.2, характеризующийся тем, что определение видовой принадлежности проводят одновременно в более чем одном образце.
| PECCHIA, SUSANNA et al | |||
| Molecular Detection of the Seed-Borne Pathogen Colletotrichum Lupini Targeting the Hyper-Variable IGS Region of the Ribosomal Cluster | |||
| Топка с несколькими решетками для твердого топлива | 1918 |
|
SU8A1 |
| Способ восстановления хромовой кислоты, в частности для получения хромовых квасцов | 1921 |
|
SU7A1 |
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| Campbell N R et al | |||
| Genotyping-in-Thousands by sequencing (GT-seq): A cost effective SNP genotyping method based | |||
Авторы
Даты
2023-11-14—Публикация
2022-12-19—Подача