Изобретение отноитя к молекулярной биологии, микробиологии и медицине и обеспечивает способ определения генотипа возбудителя гонококковой инфекции на основе анализа однонуклеотидных полиморфизмов генов «домашнего хозяйства» с использованием биологического микрочипа.
Уровень техники
Гонорея - это инфекционное заболевание, передающееся половым путем, которое признано Всемирной организацией здравоохранения (ВОЗ) серьезной угрозой репродуктивному здоровью населения. Возбудителем гонококковой инфекции являются грамотрицательные бактерии Neisseria gonorrhoeae, которые характеризуются высокой скоростью накопления как фенотипических, так и генетических адаптивных изменений. Формирование устойчивости патогенного микроорганизма N. gonorrhoeae к антибиотикам является глобальной проблемой. Зарегистрирован рост мультирезистентных штаммов N. gonorrhoeae, демонстрирующих устойчивость ко многим антимикробным препаратам, в том числе применяемым в настоящее время для терапии гонококковой инфекции цефтриаксону и азитромицину (Unemo М. and Shafer W.M. "Antimicrobial resistance in Neisseria gonorrhoeae in the 21st century: past, evolution, and future"; Clin Microbiol Rev. 2014, v. 27 (3), p. 587-613, doi:10.1128/CMR.00010-14) (Sanchez-Buso L., Cole M.J. et al. "Europe-wide expansion and eradication of multidrug-resistant Neisseria gonorrhoeae lineages: a genomic surveillance study"; Lancet Microbe. 2022, v. 3 (6), p.e452-e463, doi:10.1016/S2666-5247(22)00044-1). Для контроля за распространением антибиотикорезистентных клонов N. gonorrhoeae многими странами инициированы программы мониторинга, одним из инструментов которого является молекулярное типирование, направленное на идентификацию генетических вариантов (генотипов, сиквенс-типов, ST) возбудителя. Установление генотипа изолята позволяет анализировать пути передачи гонококковой инфекции, а также идентифицировать и контролировать распространение наиболее эпидемиологически опасных клонов. В настоящее время для генотипирования N. gonorrhoeae применяются следующие молекулярные методы:
1) NG-MAST (Neisseria gonorrhoeae multi antigen sequence typing/ мультиантигенное сиквенс-типирование N. gonorrhoeae) основан на определении нуклеотидной последовательности двух гипервариабельных локусов porB (490 по., кодирует трансмембранный белок поринового канала) и (390 п. о., кодирует β-субъединицу трансферрин-связывающего белка) (Martin I.М.,Ison С.А. et al. "Rapid sequence-based identification of gonococcal transmission clusters in a large metropolitan area"; J Infect Dis. 2004, v. 189 (8), p. 1497-1505, doi:10.1086/383047). Создана и поддерживается мировая база данных NG-MAST V2.0 (https://pubmlst.org/databases), в которую депонируются последовательности геномных локусов porB и tbpB, позволяющие определить сиквенс-тип штамма. На 2023 год база насчитывает порядка 13 тысяч аллелей porB и 3-х тысяч аллелей tbpB и постоянно дополняется новыми последовательностями. Генотипирование N. gonorrhoeae по проколу NG-MAST получило широкое распространение, поскольку NG-MAST типы сцеплены с аллельными вариантами генов, которые ассоциированы с устойчивостью к антимикробным препаратам. Установлено, что изоляты N. gonorrhoeae со сниженной чувствительностью к цефалоспоринам III поколения зачастую относятся к NG-MAST 1407, или близких к нему (Sanchez-Buso L., Cole M.J. et al. "Europe-wide expansion and eradication of multidrug-resistant Neisseria gonorrhoeae lineages: a genomic surveillance study"; Lancet Microbe. 2022, v. 3 (6), p.e452-e463, doi:10.1016/S2666-5247(22)00044-1). Обнаружено, что фенотипически устойчивые изоляты N. gonorrhoeae к макролидам (азитромицину) ассоциированы с NG-MAST типом 12302, который в течение последних лет преобладает на территории стран Евросоюза. Позднее изоляты N. gonorrhoeae из NG-MAST геногруппы 12302 были обнаружены на территории других стран, например России, что повлекло за собой резкое повышение доли устойчивых к азитромицину изолятов (Kandinov I, Dementieva Е. et al. "Emergence of Azithromycin-Resistant Neisseria gonorrhoeae Isolates Belonging to the NG-MAST Genogroup 12302 in Russia"; Microorganisms. 2023, v. 11 (5), p.doi:10.3390/microorganisms 11051226). Таким образом, генотипирование по протоколу NG-MAST позволяет отслеживать глобальные популяционно-генетические тенденции, а также особенности генетической структуры популяции N. gonorrhoeae.
Генотипирование изолятов N. gonorrhoeae по проколу NG-MAST традиционно проводят методом секвенирования по Сэнгеру фрагментов двух гипервариабельных генов porB и tbpB. Очевидно, что из-за высокой вариабельности выбранных генов, в современной популяции N. gonorrhoeae встречается большое количество новых аллелей, что приводит к появлению новых генотипов, анализ которых требует больше времени и ресурсов. Такой подход, очевидно, является трудоемким и дорогостоящим для рутинного генотипирования изолятов N. gonorrhoeae.
2) MLST (Multilocus sequence typing) (мультилокусное типирование последовательности) (Maiden M.C., Jansen van Rensburg M.J. et al. "MLST revisited: the gene-by-gene approach to bacterial genomics"; Nat Rev Microbiol. 2013, v. 11 (10), p. 728-736, doi: 10.1038/nrmicro3093).
Генотипирование по протоколу MLST основано на определении нуклеотидных последовательностей семи генов «домашнего хозяйства»: abcZ (кодирует ABC транспортер), adk (кодирует аденилаткиназу), aroE (кодирует шикиматдегидрогеназу), fumC (кодирует фумаратгидратазу), gdh (кодирует глюкозо-6-фосфатдегидрогеназу), pdhC (кодирует субъединицу пируватдегидрогеназы), pgm (кодирует фосфоглюкомутазу). В отличие от генов, последовательности которых исследуются в протоколе NG-MAST, в основе данного метода лежит анализ генов, характеризующихся низкой скоростью накопления мутаций. Последовательности и аллели данных генов, а также MLST генотипы индивидуальных изолятов N. gonorrhoeae также представлены в стандартизированной форме в общедоступной мировой базе данных (http://pubmlst.org/databases), что открывает широкие возможности для изучения генетического разнообразия различных популяций N. gonorrhoeae.
Генотипирование N. gonorrhoeae по проколу MLST позволяет выделять большое количество генетических вариантов в популяции гонококка, а также выявлять эволюционно успешные генотипы. Как и NG-MAST, определенные MLST генотипы зачастую ассоциированы с устойчивостью к антимикробным препаратам различных классов. Так, доминирующий в мире MLST сиквенс-тип 1901, ассоциирован со снижением чувствительности к цефалоспоринам III поколения (Unemo М., Golparian D. et al. "High-level cefixime- and ceftriaxone-resistant Neisseria gonorrhoeae in France: novel penA mosaic allele in a successful international clone causes treatment failure"; Antimicrob Agents Chemother. 2012, v. 56 (3), p. 1273-1280, doi:10.1128/AAC.05760-11). Также с каждым годом увеличивается количество изолятов сиквенс-типа 9363, которые ассоциированы с устойчивостью к макролидам, что, в целом, описывает мировую тенденцию к увеличению числа резистентных к антимикробным препаратам изолятов (Schaeffer J., Lippert К. et al. "Association of Phylogenomic Relatedness among Neisseria gonorrhoeae Strains with Antimicrobial Resistance, Austria, 2016-2020"; Emerg Infect Dis. 2022, v. 28 (8), p. 1694-1698, doi:10.3201/eid2808.220071).
Генотипирование изолятов N. gonorrhoeae по проколу MLST традиционно проводят методом секвенирования по Сэнгеру фрагментов семи генов abcZ, adk, aroE, fumC, gdh, pdhC, pgm. Процедура включает раздельную амплификацию каждого фрагмента с последующей очисткой ПЦР-продуктов, проведением реакции секвенирования и определением последовательности отдельного локуса с использованием капиллярного секвенатора. Такой подход, очевидно, является трудоемким и дорогостоящим для рутинного генотипирования изолятов N. gonorrhoeae.
Описан метод генотипирования N. gonorrhoeae по проколу MLST с помощью ПЦР в реальном времени посредством анализа 14 информативных однонуклеотидных полиморфизмов в вышеперечисленных генах (Whiley D.M., Goire N. et al. "Real-time PCR genotyping of Neisseria gonorrhoeae isolates using 14 informative single nucleotide polymorphisms on gonococcal housekeeping genes"; J Antimicrob Chemother. 2013, v. 68 (2), p. 322-328, doi:10.1093/jac/dks381). С помощью набора праймеров и специфичных флуоресцентно-меченных зондов проводят амплификацию фрагментов генов abcZ, adk, aroE, fumC, gdh, pdhC, pgm и регистрируют флуоресцентные сигналы в зависимости от полиморфизма в исследуемом локусе. Для каждого изолята одновременно используется не менее 19 реакционных объемов, в каждом из которых определяют только один информативный полиморфизм. По завершении амплификации для изолята получают профиль, состоящий из последовательности информативных однонуклеотидных полиморфизмов, который соотносят с подобными записями в базе данных (https://pubmlst.org/databases). По результатам анализа изолят относят к уже известному MLST генотипу, обладающему определенным профилем, либо присваивают новый MLST генотип, имеющий уникальный, ранее не встречавшийся в базе профиль. Таким образом, путем определения нескольких однонуклеотидных полиморфизмов в семи генах «домашнего хозяйства)) N. gonorrhoeae, можно предсказать MLST генотип с высокой (до 80%) точностью, что избавляет от необходимости секвенировать локусы семи генов. К недостаткам данного метода можно отнести необходимость использования для каждого изолята до 19 индивидуальных реакционных объемов с целью определения необходимых информативных однонуклеотидных полиморфизмов, что позволяет считать процедуру трудозатратной.
3) cgMLST (core genome multi-locus sequence typing) (мультилокусное типирование последовательности по кор-геному).
Новые подходы к молекулярной эпидемиологии гонококковой инфекции основаны на использовании технологий высокопроизводительного секвенирования (NGS), позволяющих одновременно оценивать как совокупность генов, характеризующих происхождение анализируемого клинического изолята, так и совокупность имеющихся у него генетических детерминант антибиотикорезистентности (Harrison О.В., Clemence М. et al. "Genomic analyses of Neisseria gonorrhoeae reveal an association of the gonococcal genetic island with antimicrobial resistance"; J Infect. 2016, v. 73 (6), p. 578-587, doi:10.1016/j.jinf.2016.08.010). По сути, NGS позволяет решать задачи генотипирования, в частности, по протоколам NG- MAST и MLST, на более высоком уровне, при этом прямо, а не косвенно связывая генотип изолята и фенотипическую чувствительность анализируемых клинических изолятов (Harrison О.B., Cehovin A. et al. "Neisseria gonorrhoeae Population Genomics: Use of the Gonococcal Core Genome to Improve Surveillance of Antimicrobial Resistance"; J Infect Dis. 2020, v. 222 (11), p. 1816-1825, doi:10.1093/infdis/jiaa002). NGS успешно используется для идентификации детерминант резистентности, исследования филогенетических взаимосвязей, популяционной структуры и молекулярной эпидемиологии изолятов (Golparian D., Dona V. et al. "Antimicrobial resistance prediction and phylogenetic analysis of Neisseria gonorrhoeae isolates using the Oxford Nanopore MiniON sequencer"; Sci Rep.2018, v. 8 (1), p. 17596, doi:10.1038/s41598-018-35750-4).
Для молекулярного генотипирования штаммов N. gonorrhoeae с использованием технологий NGS разработано мультилокусное генотипирование на основе кор-генома cgMLST. Для генотипирования изолятов N. gonorrhoeae по схеме cgMLST используется 1659 генов как хромосомной, так и плазмидной локализации. В схему типирования cgMLST также включены гены, замены в которых ассоциированы с резистентностью к антимикробным препаратам различных классов. Несмотря на колоссальную информативность, методу присущи недостатки, такие как невысокая дискриминирующая способность в отношении отдельных нуклеотидов, требующая большого покрытия генома и, соответственно, дорогостоящих наборов реагентов, недостаточно валидированные биоинформатические инструменты обработки данных и интерпретации результатов (пайплайны). Таким образом, метод cgMLST пока не может в полной мере заместить в рутинной практике протоколы NG-MAST и MLST.
4) NG-STAR (Neisseria gonorrhoeae Sequence Typing for Antimicrobial Resistance) (Генотипирование Neisseria gonorrhoeae с анализом детерминант резистентности к АМП) (Golparian D., Sanchez-Buso L. et al. "Neisseria gonorrhoeae Sequence Typing for Antimicrobial Resistance (NG-STAR) clonal complexes are consistent with genomic phylogeny and provide simple nomenclature, rapid visualization and antimicrobial resistance (AMR) lineage predictions"; J Antimicrob Chemother. 2021, v. 76 (4), p. 940-944, doi:10.1093/jac/dkaa552). Генотипирование изолятов N. gonorrhoeae с одновременным анализом детерминант резистентности (NG-STAR) - стандартизированный метод классификации семи хорошо охарактеризованных генов Ж gonorrhoeae (penA, mtrR, ponA, gyrA, parC и 23S pPHK), ассоциированных с устойчивостью к антимикробным препаратам трех классов, включая цефалоспорины, макролиды и фторхинолоны. Каждой последовательности присваивается номер аллели согласно специально разработанной базе данных открытого пользования (https://ngstar.canada.ca/). Комбинация аллелей семи генов позволяет определить генотип изолята по протоколу NG-STAR, а также предсказать профиль фенотипической чувствительности к антибиотикам на основе уже известных генотипов. База данных NG-STAR содержит около 2-х тысяч генетических профилей, соотнесенные с данными по чувствительности к указанным выше антибактериальным препаратам.
Определение уровней устойчивости N. gonorrhoeae к антимикробным препаратам по протоколу NG-STAR является достоверным (95% соответствий с экспериментально определенными значениями минимальных подавляющих концентраций (МПК) к антибиотикам) (Demczuk W., Sidhu S. et al. "Neisseria gonorrhoeae Sequence Typing for Antimicrobial Resistance, a Novel Antimicrobial Resistance Multilocus Typing Scheme for Tracking Global Dissemination of N. gonorrhoeae Strains"; J Clin Microbiol. 2017, v. 55 (5), p.1454-1468, doi:10.1128/JCM.00100-17)). Вместе с тем, данный метод генотипирования не подходит для изучения молекулярной эпидемиологии гонококковой инфекции, поскольку в большинстве случаев N. gonorrhoeae распространяется по сети контактов через клональное размножение. Таким образом, в разных популяциях зачастую присутствует большое количество идентичных NG-STAR генотипов, что не позволяет в полной мере выделять и контролировать распространение наиболее эпидемиологически значимых клонов. Также процедура анализа, реализованная в методе, предполагает установление нуклеотидной последовательности семи локусов, что является трудоемкой задачей и ограничивает его применение в рутинной практике.
Таким образом, из анализа уровня техники следует, что в настоящее время в данной области существует острая потребность в разработке способа генотипирования изолятов N. gonorrhoeae, который бы выгодно отличался от известных из уровня техники решений простотой проведения анализа, высокой специфичностью и информативностью в отношении идентифицируемых генотипов, а также невысокой стоимостью.
Раскрытие сущности изобретения
Техническим результатом изобретения является создание способа генотипирования изолятов N. gonorrhoeae посредством идентификации информативных однонуклеотидных полиморфизмов в генах «домашнего хозяйства» с помощью биологического микрочипа.
В заявленном способе предложено использование мультиплексной амплификации и флуоресцентного маркирования фрагментов генома N. gonorrhoeae с получением флуоресцентно-меченных ПЦР-продуктов, при этом в качестве матрицы может быть использована геномная ДНК, выделенная из клинических изолятов - культур клеток гонококка, выращенных в жидкой или на твердой средах. Заявленный способ также предусматривает использование оригинального биологического микрочипа с иммобилизованными специфическими олигонуклеотидными зондами, процедуры гибридизации, регистрации и интерпретации результатов.
В своем первом аспекте данное изобретение обеспечивает способ генотипирования изолятов N. gonorrhoeae посредством идентификации информативных однонуклеотидных полиморфизмов в генах «домашнего хозяйства», с использованием биологического микрочипа. Способ настоящего изобретения основан на двухстадийной ПЦР с получением флуоресцентно меченных одноцепочечных фрагментов с последующей гибридизацией этих фрагментов на биологическом микрочипе, регистрацию и интерпретацию результатов гибридизации. Способ предусматривает следующие стадии:
а) первый этап мультиплексной ПЦР при использовании геномной ДНК изолята N. gonorrhoeae в качестве матрицы и первого набора специфичных праймеров для амплификации фрагментов генов abcZ, adk, aroE, fumC, gdh,pdhC, pgm;
б) второй этап мультиплексной ПЦР, проводимой по асимметричному типу, с использованием в качестве матрицы ПЦР-продуктов, полученных на стадии (а), второго набора специфичных праймеров, а также смеси четырех дезоксинуклеозидтрифосфатов, в которой один из четырех дезоксинуклеозидтрифосфатов является флуоресцентно меченым, что позволяет получать преимущественно одноцепочечные флуоресцентно меченые фрагменты генов abcZ, adk, aroE, fumC, gdh, pdhC, pgm, последовательности которых содержат информативные однонуклеотидные полиморфизмы;
в) обеспечение биологического микрочипа для идентификации информативных однонуклеотидных полиморфизмов, представляющего собой подложку, содержащую множество дискретных элементов, в каждом из которых иммобилизован уникальный олигонуклеотидный зонд, имеющий последовательность, комплементарную последовательности одноцепочечного фрагмента, полученного на стадии (б) и выбранную из группы, включающей: i) последовательности фрагмента гена abcZ, содержащего информативные однонуклеотидные полиморфизмы в положениях 34 и 205; ii) последовательности фрагмента гена adk, содержащего информативные однонуклеотидные полиморфизмы в положениях 565 и 611; iii) последовательности фрагмента гена aroE, содержащего информативные однонуклеотидные полиморфизмы в положениях 1112 и 1271; iv) последовательности фрагмента гена fumC, содержащего информативные однонуклеотидные полиморфизмы в положениях 1526, 1646, 1649, 1685 и 1691; v) последовательности фрагмента гена gdh, содержащего информативные однонуклеотидные полиморфизмы в положениях 1886, 1937, 2028, 2282; vi) последовательности фрагмента гена pdhC, содержащего информативные однонуклеотидные полиморфизмы в положениях 2513, 2775; vii) последовательности фрагмента гена pgm, содержащего информативные однонуклеотидные полиморфизмы в положении 3276;
г) гибридизацию амплифицированных флуоресцентно-меченных ПЦР-продуктов, полученных на стадии (б), на биологическом микрочипе, полученном на стадии (в), с образованием дуплексов с иммобилизованными зондами в условиях, обеспечивающих разрешение в один нуклеотид между образующимися в результате гибридизации совершенными и несовершенными дуплексами;
д) регистрацию и интерпретацию результатов гибридизации на биологическом микрочипе, проведенной на стадии (г), с использованием анализатора флуоресценции и программного обеспечения.
В одном из своих воплощений способ характеризуется тем, что на стадии (а) используют первый набор специфичных праймеров, последовательности которых представлены SEQ ID NO: 1-14.
В еще одном из воплощений способ характеризуется тем, что на стадии (б) используют второй набор специфичных праймеров, последовательности которых представлены SEQ ID NO: 15- 28.
В своем следующем воплощении способ характеризуется тем, что на стадии (б) обратные праймеры используют, по меньшей мере, в десятикратном молярном избытке по отношению к прямым праймерам, что позволяет получать преимущественно одноцепочечные ПЦР продукты.
В своем следующем воплощении способ характеризуется тем, что на стадии (б) в качестве флуоресцентно меченого дезоксинуклеозидтрифосфата используют флуоресцентно меченый дезоксиуридинтрифосфат.
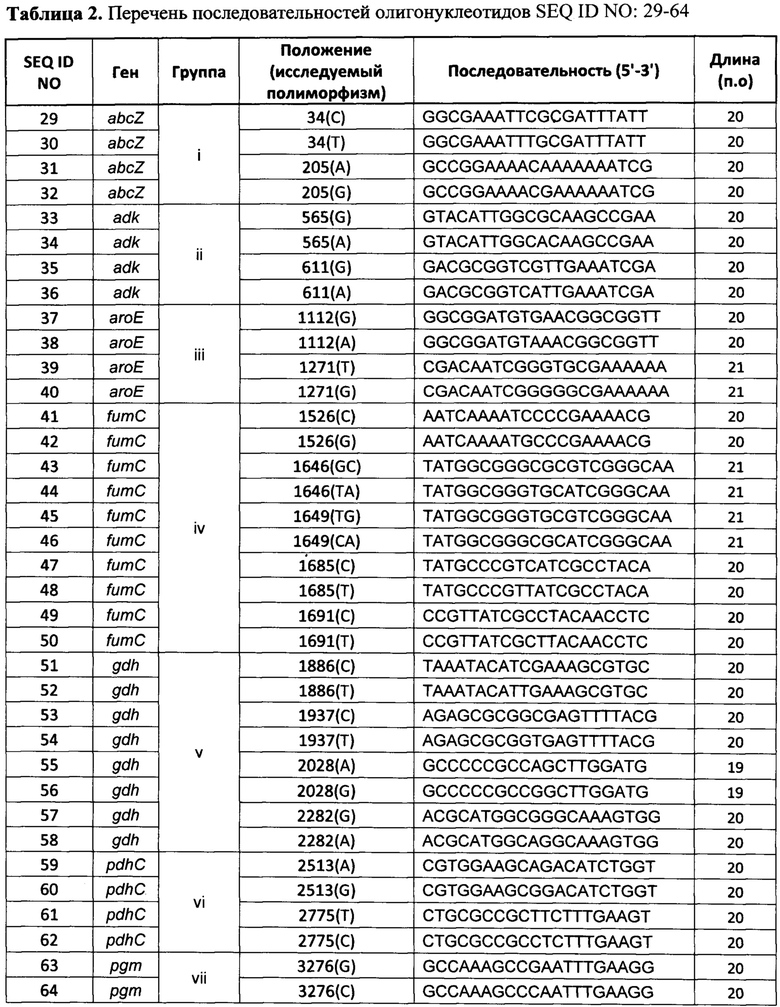
В еще одном из воплощений способ характеризуется тем, что биологический микрочип представляет собой подложку с гидрогелевыми элементами, полученными химическим способом или методом фотоиндуцируемой сополимеризации и содержащими иммобилизованные олигонуклеотидные зонды, последовательности которых представлены SEQ ID NO: 29-64.
В своем следующем воплощении способ характеризуется тем, что на стадии (д) регистрируют максимальный сигнал в каждой из групп, отвечающий образованию совершенного гибридизационного дуплекса, и устанавливают информативные однонуклеотидные полиморфизмы в каждом из анализируемых положений генов abcZ, adk, aroE,fumC,gdh,pdhC,pgm.
В следующем воплощении способ характеризуется тем, что на стадии (д) при интерпретации результатов получают уникальный нуклеотидный профиль, состоящий из последовательности информативных однонуклеотидных полиморфизмов, и устанавливают генотип изолята посредством сравнения полученного профиля с эталонными профилями из базы данных pubMLST.
Другие аспекты настоящего изобретения будут ясны из прилагаемых фигур, подробного описания и формулы изобретения.
Краткое описание фигур и таблиц
Фигура 1. Схема размещения элементов биологического микрочипа для идентификации информативных однонуклеотидых полиморфизмов в семи генах «домашнего хозяйства» N. gonorrhoeae. Элементы биологического микрочипа представлены в виде кругов. Обозначение элемента - анализируемые однонуклеотидные полиморфизмы (SNP) и их положения, которые имеют единую последовательную систему нумераций для всех анализируемых локусов: ген abcZ (положения 34, 205), adk (положения 565, 611), aroE (положения 1112, 1271), fumC (положения 1526, 1646, 1649, 1685, 1691), gah (положения 1886, 1937, 2028, 2282),pdhC (положения 2513, 2775), pgm (положение 3276). На схеме разными цветами выделены группы зондов (i-vii), которые соответствуют каждому гену, и внутри которых на стадии регистрации и интерпретации результатов гибридизации выбирается максимальный флуоресцентный сигнал и идентифицируется информативный однонуклеотидный полиморфизм в каждом положении. М - элементы, содержащие флуоресцентный маркер для обработки сигналов элементов биологического микрочипа.
Фигура 2. Гибридизационная картина (флуоресцентное изображение) биологического микрочипа и нуклеотидный профиль при анализе ДНК изолята N. gonorrhoeae, имеющего MLST генотип 1594.
Фигура 3. Гибридизационная картина (флуоресцентное изображение) биологического микрочипа и нуклеотидный профиль при анализе ДНК изолята N. gonorrhoeae, имеющего MLST генотип 1901.
Фигура 4. Гибридизационная картина (флуоресцентное изображение) биологического микрочипа и нуклеотидный профиль при анализе ДНК изолята N. gonorrhoeae, имеющего MLST генотип 9363.
Таблица 1. Перечень последовательностей и концентраций праймеров SEQ ID NO: 1-28, используемых в реакционных смесях на этапах мультиплексной ПЦР.
Таблица 2. Перечень последовательностей олигонуклеотидов SEQ ID NO: 29-64, иммобилизованных на биологическом микрочипе.
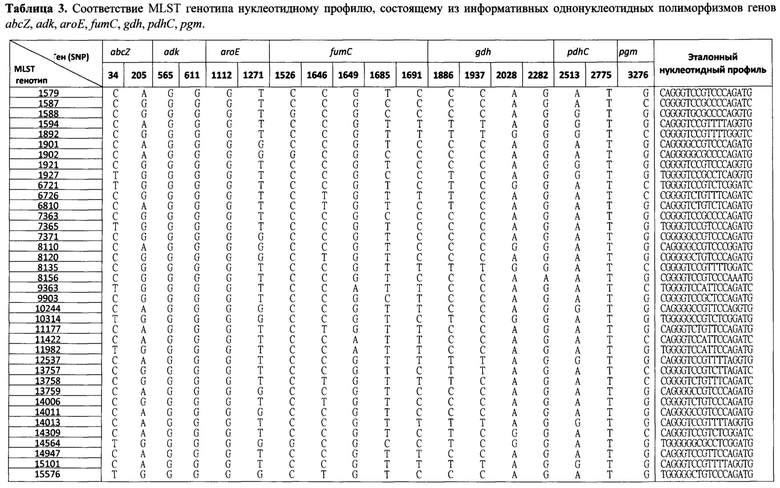
Таблица 3. Соответствие MLST генотипа нуклеотидному профилю, состоящему из информативных однонуклеотидных полиморфизмов генов abcZ, adk, aroE, fumC, gdh, pdhC, pgm. Перечень эталонных MLST генотипов и соответствующие им нуклеотидные профили из базы данных pubMLST.
Осуществление изобретения
В результате проведенных обширных научных исследований, авторы настоящего изобретения обнаружили, что задача разработки способа генотипирования изолятов N. gonorrhoeae может быть успешно решена путем использования биологических микрочипов (биочипов), содержащих олигонуклеотидные зонды, последовательности которых специфичны к локусам, содержащие информативные однонуклеотидные полиморфизмы в семи генах «домашнего хозяйства" возбудителя. Технология гидрогелевых биологических микрочипов низкой плотности успешно зарекомендовала себя в фундаментальных биомедицинских исследованиях и клинической лабораторной диагностике (Gryadunov D.A., Shaskolskiy B.L. et al. "The EIMB Hydrogel Microarray Technology: Thirty Years Later"; Acta Naturae. 2018, v. 10 (4), p. 4-18).
Заявляемый в настоящем изобретении способ генотипирования N. gonorrhoeae на основе технологии биологических микрочипов низкой плотности выгодно отличается от известных из уровня техники методов возможностью одновременной идентификации 18 информативных однонуклеотидных полиморфизмов с целью установления MLST генотипа изолята N. gonorrhoeae. Метод не требует дорогостоящего оборудования и высококвалифицированного персонала. Данные, полученные с помощью заявленного способа, могут быть использованы для эпидемиологических исследований, и мониторинга циркулирующих изолятов на территории России и других стран, включая выявление событий трансграничного переноса возбудителя.
Образцы клинических изолятов (культуры клеток) N. gonorrhoeae подвергают процедурам лизиса и очистки ДНК от клеточного дебриса с целью получения геномной ДНК, пригодной для проведения ПЦР. Выделение ДНК N. gonorrhoeae может быть проведено с помощью известных в данной области способов (например, Kalia A., Rattan А. et al. "A method for extraction of high-quality and high-quantity genomic DNA generally applicable to pathogenic bacteria"; Anal Biochem. 1999, v. 275 (1), p.1-5, doi:10.1006/abio.1999.4259), или любого специализированного коммерчески доступного набора реагентов для выделения ДНК из культуры клеток, например «Набор реагентов для выделения нуклеиновых кислот «ПРОБА-НК» (ООО «НПО ДНК-Технология», Россия), или «Набор реагентов для выделения ДНК бактерий и грибов из культур микроорганизмов для диагностики in vitro (ПРОБА-КМ)» (ООО «НПО ДНК-Технология», Россия). Полученный препарат геномной ДНК N. gonorrhoeae используют в качестве матрицы для двухстадийной мультиплексной ПЦР.
При выборе мишеней для амплификации используют данные полногеномного секвенирования клинических образцов из открытых баз данных, предпочтительно pubMLST (https://pubmlst.org/databases). Применяя специализированное программное обеспечение, например Bioedit (Ibis biosciences, США) или Ugene v. 44 (Unipro, Россия), или другие коммерчески доступные программы, или программы, свободно доступные в сети Internet, проводят множественное выравнивание семи генов «домашнего хозяйства», являющимися одними из наиболее консервативных в геноме N. gonorrhoeae. Выровненные последовательности генов объединяют в единую линейную последовательность длиной 3286 пар оснований строго в порядке встречаемости в хромосоме и выбирают однонуклеотидные полиморфизмы, позволяющие дифференцировать существующие MLST генотипы N. gonorrhoeae. Предпочтительно, чтобы локусы, подлежащие амплификации, содержали наиболее полиморфные положения, как описано ранее (Bennett J.S., Jolley K.А. et al. "Species status of Neisseria gonorrhoeae: evolutionary and epidemiological inferences from multilocus sequence typing"; BMC Biol. 2007, v. 5 p.35, doi:10.1186/1741-7007-5-35).
Мультиплексную амплификацию семи фрагментов генов «домашнего хозяйства)), содержащих информативные однонуклеотидные полиморфизмы, проводят в две стадии:
на стадии (а) в качестве матрицы для амплификации используют препараты геномной ДНК N. gonorrhoeae и первый набор специфичных праймеров для амплификации фрагментов генов «домашнего хозяйства». В ходе амплификации происходит одновременная наработка семи фрагментов генома. Праймеры для проведения мультиплексной ПЦР выбирают таким образом, чтобы они фланкировали локусы, содержащие информативные однонуклеотидные полиморфизмы. Используя специализированное программное обеспечение, например Oligo v. 6.3 (Molecular Biology Insights Inc., США) или Fast PCR (http://www.biocenter.helsinki.fi/bi/ Programs/fastpcr.htm) или другие коммерчески доступные программы, или программы, свободно доступные в сети Internet, рассчитывают температуры плавления праймеров и, варьируя их длину, добивались того, чтобы разброс температур отжига праймеров внутри набора не превышал 3-4°С. При подборе праймеров избегают таких последовательностей, которые способны формировать вторичные структуры типа шпильки с высокими температурами плавления, а также последовательностей, образующих между собой дуплексы, состоящие более чем из трех-пяти нуклеотидов. Каждый выбранный праймер должен обладать уникальной специфичностью в отношении анализируемого участка последовательности генома N. gonorrhoeae Специфичность праймеров проверяют с помощью программного обеспечения, использующего поиск в базах нуклеотидных последовательностей по алгоритму BLAST (например, www.ncbi.nlm.nih.gov/BLAST):
на стадии (б) в качестве матрицы для амплификации используют ПЦР-продукты, полученные на стадии (а), и второй набор праймеров, последовательности которых специфичны к последовательностям фрагментов генов «домашнего хозяйства», содержащих информативные однонуклеотидные полиморфизмы. Праймеры для проведения второй стадии амплификации выбирают с учетом требований, изложенных выше, с тем отличием, что по меньшей мере один из праймеров выбирается внутри ПЦР-фрагмента, получаемого на первой стадии, что повышает специфичность реакции. Праймеры выбирают таким образом, чтобы размер амплифицируемых фрагментов на второй стадии составлял 70-400 п.o. Большая длина ПЦР-продуктов, получаемых на второй стадии, затрудняет эффективную диффузию анализируемых фрагментов генома в гелевых элементах биологического микрочипа при гибридизации, что в конечном итоге может привести к уменьшению количества образовавшихся гибридизационных дуплексов и, как следствие, падению флуоресцентного сигнала в ячейках. При подборе праймеров следует учитывать тот факт, что на выходе реакции получаются преимущественно одноцепочечные флуоресцентно меченные продукты, комплементарные олигонуклеотидам, иммобилизованным на биологическом микрочипе. При этом обратные праймеры используют, по меньшей мере, в десятикратном молярном избытке по отношению к прямым праймерам, в качестве флуоресцентно меченого дезоксинуклеозидтрифосфата добавляют флуоресцентно меченый дезоксиуридинтрифосфат (dUTP), что позволяет получать преимущественно одноцепочечные флуоресцентно-меченные ПЦР продукты.
В качестве флуоресцентного красителя может быть использован любой флуоресцентный краситель, который может быть химически включен в молекулу дезоксинуклеозидтрифосфата таким образом, чтобы не препятствовать в существенной степени прохождению полимеразной цепной реакции и последующей гибридизации полинуклеотидной молекулы, содержащей такие флуоресцентно меченые нуклеотидные остатки, с иммобилизованными олигонуклеотидными зондами. В случае флуоресцентно меченого дезоксиуридинтрифосфата, например, флуоресцентный краситель может быть присоединен к 5'-концу аминоаллильного производного dUTP. Примеры таких красителей хорошо известны специалисту в данной области техники и включают красители флуоресцеинового (TAMRA®, ROX®, JOE®), родаминового (Texas Red®), полиметинового (Су3®, Су5®, Су5.5®, Су7®) рядов (Ranasinghe R. and Brown Т. Fluorescence based strategies for genetic analysis. Chem. Commun., 2005, 5487-5502). Наиболее предпочтительными являются красители, спектр возбуждения которых лежит в длинноволновой (красной) области спектра, что позволяет использовать для возбуждения флуоресценции недорогие источники возбуждающего излучения типа полупроводниковых лазеров. В частности, используют флуоресцентные красители с длиной волны возбуждения, равной (640±5) нм и длиной волны флуоресценции, равной (665±5) нм от компании «Люмипроб РУС» (Москва, Россия).
Заявленный способ также предусматривает использование оригинального биологического микрочипа с иммобилизованными олигонуклеотидными зондами, последовательности которых комплементарны локусам N. gonorrhoeae, содержащим информативные однонуклеотидные полиморфизмы.
Выбор олигонуклеотидных зондов проводят с учетом длины и сложности анализируемой последовательности, в частности, наличие повторов и протяженных гомополимерных последовательностей для обеспечения специфичности зондов в отношении анализируемой последовательности. Для каждой позиции, для которой анализируются информативные однонуклеотидные полиморфизмы, подбирают набор специфичных олигонуклеотидов, способный выявлять известные варианты замен. С использованием программного обеспечения Oligo v. 6.3 (Molecular Biology Insights Inc., США), рассчитывают температуры плавления олигонуклеотидов и, варьируя их длину, добиваются того, чтобы разброс температур плавления составлял не более 2-3°С. Избегают олигонуклеотидов, которые способны формировать вторичные структуры типа «шпильки» с высокими температурами плавления. Положение определяемых информативных однонуклеотидных полиморфизмов выбирают по возможности не далее 1 -4 нуклеотида от середины соответствующего дискриминирующего олигонуклеотида.
Биологический микрочип может представлять собой матрицу, состоящую из гидрогелевых элементов полусферической формы («трехмерный» биологический микрочип), содержащих ковалентно иммобилизованные олигонуклеотиды, изготовленный, например, методом сополимеризационной иммобилизации, разработанной и запатентованный Институтом молекулярной биологии им. В.A. Энгельгардта Российской академии наук (ИМБ РАН) (Gryadunov D.A., Shaskolskiy B.L. et al. "The EIMB Hydrogel Microarray Technology: Thirty Years Later"; Acta Naturae. 2018, v. 10 (4), p. 4-18). При изготовлении гидрогелевого биологического микрочипа предварительно формируют композиции, содержащие гелеобразующие мономеры и олигонуклеотидные зонды, подлежащие иммобилизации. Композиции наносят на подложки из стекла или пластика в виде массива микрокапель с помощью автоматического микродозатора (робота). Под действием УФ-излучения происходит полимеризация геля с одновременной ковалентной иммобилизацией зондов в элементах - каплях объемом до 0,1 нл.
Заявленное изобретение также включает гибридизацию флуоресцентно-меченных продуктов на биологическом микрочипе, с образованием совершенных и несовершенных дуплексов с иммобилизованными олигонуклеотидными зондами. Гибридизацию проводят в растворе, содержащем буферный компонент для поддержания рН, соль для создания ионной силы и хаотропный (дестабилизирующий водородные связи) агент, при температуре, зависящей от температуры плавления иммобилизованных на микрочипе олигонуклеотидов, например, как описано в работе (Mikhailovich V.M., Lapa S.A. et al. "Detection of rifampicin-resistant Mycobacterium tuberculosis strains by hybridization and polymerase chain reaction on a specialized ТВ-microchip"; Bull Exp Biol Med. 2001, v. 131 (1), p.94-98, doi:10.1023/a:1017555318388).
Дискриминацию совершенных и несовершенных дуплексов выполняют путем сравнения интенсивностей флуоресценции ячеек, в которых образовались дуплексы. Интенсивность сигнала в ячейке, в которой образуется совершенный гибридизационный дуплекс (Icoв) выше, чем в таковой, где образовался несовершенный дуплекс (Iнесов). Проведение гибридизации при оптимальных условиях (температура, подобранная концентрация хаотропного агента и ионная сила гибридизационного буфера) позволяют добиться соотношения Icoв/Iнесов≥2 между двумя ячейками, содержащими зонды, принадлежащие одной группе, и различающиеся на один нуклеотид.
Заявленное изобретение также включает получение уникального нуклеотидного профиля, состоящего из последовательности информативных однонуклеотидных полиморфизмов, и установление генотипа изолята посредством сравнения полученного профиля с эталонными профилями из базы данных pubMLST. В каждой из групп элементов биологического микрочипа, соответствующих отдельному информативному однонуклеотидному полиморфизму, регистрируют максимальный сигнал, отвечающий совершенному гибридизационному дуплексу. Тем самым, по результатам анализа геномной ДНК изолята N. gonorrhoeae на биологическом микрочипе получают набор (последовательность) информативных олигонуклеотидных полиморфизмов, из которых составляют нуклеотидный профиль. Полученный профиль сравнивают с эталонными генотип-специфичными профилями из базы данных pubMLST по принципу максимального совпадения и устанавливают генотип изолята. Алгоритм регистрации и интерпретации результатов гибридизации на биологическом микрочипе с определением набора (последовательности) информативных однонуклеотидных полимфорфизмов и выдачей информации о генотипе изолята может быть реализован в программном обеспечении анализатора флуоресценции биочипов.
Далее изобретение будет проиллюстрировано примерами, которые предназначены для обеспечения лучшего понимания сущности заявленного изобретения, но не должны рассматриваться как ограничивающие данное изобретение.
Пример. Генотипирование изолятов N. gonorrhoeae посредством идентификации информативных однонуклеотидных полиморфизмов в генах «домашнего хозяйства» методом мультиплексной ПЦР с последующей гибридизацией на гидрогелевом биочипе, регистрацией и интерпретацией результатов.
I. Получение геномной ДНК изолята N. gonorrhoeae
При осуществлении изобретения используют три клинических изолята из коллекции Государственного научного центра дерматовенерологии и косметологии Минздрава России (Kubanov A., Solomka V. et al. "Summary and Trends of the Russian Gonococcal Antimicrobial Surveillance Programme, 2005 to 2016"; J Clin Microbiol. 2019, v. 57 (6), p.doi:10.1128/JCM.02024-18), для которых ранее референсным методом были определены генотипы согласно протоколу MLST: первый изолят имел MLST генотип 1594; второй -MLST генотип 1901; третий - MLST генотип 9363.
Клетки изолятов высевают на шоколадном агаре GC (Difco, США) с добавлением ростовых добавок IsoVitaleX Enrichmen (Becton Dickinson, США). Чашку Петри инкубируют в течение ночи при температуре 37°С, 5% CO2. Собирают выросшие колонии при помощи культуральной петли в 100 мкл PBS буфера. Выделение ДНК проводят с использованием экспресс-наборов для выделения ДНК НПФ «Литех» (Россия). Конечные концентрации ДНК измеряют с помощью спектрофотометра NanoDrop 2000 и Qubit 4 Fluorometer (Thermo Scientific, США), диапазон концентраций находится в пределах 20-100 нг/мкл.
II. Конструирование, синтез и очистка праймеров для амплификации генов «домашнего хозяйства» N. gonorrhoeae
Конструирование праймеров проводят в программе Bioedit (Ibis biosciences, США). В качестве референсных геномов используют геномы 14 штаммов (Unemo М., Golparian D. et al. "The novel 2016 WHO Neisseria gonorrhoeae reference strains for global quality assurance of laboratory investigations: phenotypic, genetic and reference genome characterization"; J Antimicrob Chemother. 2016, v. 71 (11), p.3096-3108, doi:10.1093jac/dkw288). Проводят множественное выравнивание последовательностей семи генов «домашнего хозяйства», являющихся одними из наиболее консервативных в геноме N. gonorrhoeae. Выровненные последовательности генов объединяют в единую линейную последовательность длиной 3286 пар оснований строго в порядке встречаемости в хромосоме и выбирают однонуклеотидные полиморфизмы, позволяющие дифференцировать существующие MLST генотипы N. gonorrhoeae:
- положения 34, 205 гена abcZ;
- положения 565, 611 гена adk;
- положения 1112,1271 гена aroE;
- положения 1526,1646,1649,1685,1691 гена fumC;
- положения 1886, 1937, 2028, 2282 гена pdh;
- положения 2513,2775 гена pdhC;
- положение 3276 гена pgm.
Праймеры для проведения мультиплексной ПЦР выбирают таким образом, чтобы они фланкировали анализируемые положения генов abcZ, adk, aroE, fumC, gdh, pdhC, pgm. Для этапа амплификации (а) конструируют последовательности семи прямых праймеров и семи обратных праймеров (Таблица 1, последовательности SEQ ID NO: 1-14). Для этапа амплификации (б) конструируют последовательности семи прямых праймеров и семи обратных праймеров (Таблица 1, последовательности SEQ ID NO: 15-28).
Праймеры получают путем стандартного фосфорамидитного синтеза со шкалой от 0,2 до 1 μМ на автоматическом синтезаторе MerMade 48Х (Biosearch Technologies, США). Очистку олигонуклеотидных праймеров проводят путем обращенно-фазовой ВЭЖХ на колонке Supelco LC-18 (5 мкм, 4,6 × 250 мм, Sigma, США) с элюцией или 0,05М ацетатом триэтиламмония (ТЕАА), рН 7,0, или градиентом концентраций CH3CN от 0 до 50% в 0,05 М ТЕАА.
III. Изготовление гидрогелевого биологического микрочипа для генотипирования изолятов N. gonorrhoeae
Олигонуклеотидные зонды с последовательностями SEQ ID NO: 29-64 (Таблица 2) получают путем стандартного фосфорамидитного синтеза со шкалой от 0,2 до 1 μМ на автоматическом синтезаторе MerMade 48Х (Biosearch Technologies, США). Модификацию аминогруппой 5'-конца выполняют с помощью 5'-Amino-Modifier С6 и Unilink™ Amino-Modifier (Glen Research, США). Очистку олигонуклеотидных зондов проводят путем обращенно-фазовой ВЭЖХ на колонке Supelco LC-18 (5 мкм, 4,6 × 250 мм, Sigma, США) с элюцией или 0,05М ацетатом триэтиламмония (ТЕАА), рН 7,0, или градиентом концентраций CH3CN от 0 до 50% в 0,05М ТЕАА.
Биологические микрочипы, содержащие олигонуклеотидные зонды, полученные как описано выше, и иммобилизованные в гидрогеле, изготавливают в соответствии с опубликованной ранее методикой (Shaskolskiy В., Kandinov I. et al. "Hydrogel Droplet Microarray for Genotyping Antimicrobial Resistance Determinants in Neisseria gonorrhoeae Isolates"; Polymers (Basel). 2021, v. 13 (22), p. doi:10.3390/polym13223889). Композиции для нанесения включают 4,75% метакриламид, 0,25% N,N'-метиленбисакриламид, 50% глицерин, N,N,N',N'-5% тетраметилендиамин (TEMED) и олигонуклеотидные зонды в концентрации 2000 пмоль/мкл. Подготовленные в пробирках композиции переносят в 384-луночный планшет, и наносят на полимерные пластины (ООО «РИ-СК», Россия) в виде массива капель-полусфер объемом ~0,1 нл с помощью автоматического микродозатора - установки для переноса гидрогелей ИМБ РАН. Диаметр полусферических гелевых ячеек составляет 100 мкм, расстояние между центрами ячеек -также 100 мкм. Полимеризацию гелевых элементов проводят под действием УФ с максимальной длиной волны 350 нм и интенсивностью 0,06 μ.W/см2 в течение 40 мин при 20°С. После полимеризации пластины промывают 0,1 М буфером PBS, содержащим 0,1% Tween 20, затем деионизированной водой и высушивают при комнатной температуре. Разрезание полимеризационных пластин на индивидуальные подложки микрочипов выполняют с использованием лазера, фиксированного на двухкоординатном позиционере, после чего подложки вкладывают в пластиковые держатели формата предметного стекла и проводят технологический контроль качества. Проверку каждого микрочипа выполняют с использованием портативного флуоресцентного микроскопа, оснащенного CCD-камерой и программным обеспечением, распознающим элементы биологического микрочипа и обсчитывающим основные параметры (диаметр, объем, отклонения от заданных геометрических параметров матрицы) с последующей статистической обработкой и выдачей информации о пригодности микрочипа для дальнейшего использования. В случае положительного заключения проводят окончательную сборку микрочипа посредством монтажа составной гибридизационной камеры объемом 30 мкл (Shaskolskiy В., Kandinov I. et al. "Hydrogel Droplet Microarray for Genotyping Antimicrobial Resistance Determinants in Neisseria gonorrhoeae Isolates"; Polymers (Basel). 2021, v. 13 (22), p.doi:10.3390/polym13223889) и используют готовый биологический микрочип для гибридизационного анализа.
Схема размещения элементов биологического микрочипа для идентификации информативных однонуклеотидых полиморфизмов в семи генах «домашнего хозяйства» N. gonorrhoeae приведена на Фигуре 1. Биологический микрочип состоит из 42 элементов, 36 из которых содержат иммобилизованные олигонуклеотидные зонды с последовательностями SEQ ID NO: 29-64 (Таблица 2), три элемента с иммобилизованным флуоресцентным маркером - аналогом красителя Су5. Ячейки с маркером (М) используются для правильного позиционирования биологического микрочипа при регистрации флуоресцентного изображения после проведения гибридизации и его обработки программным обеспечением анализатора биологических микрочипов (сканера) при автоматическом получении результатов. Также биологический микрочип содержит три ячейки гидрогеля, свободные от олигонуклеотидов и красителей (empty), предназначенные для контроля качества его отмывки.
Биологический микрочип содержит семь групп, которые соответствуют генам «домашнего хозяйства» N. gonorrhoeae что, обеспечивает определение информативных полиморфизмов в гене abcZ - группа i (положения 34, 205), гене adk - группа ii (положения 565, 611), гене aroE - группа iii (положения 1112, 1271), гене fumC - группа iv (положения 1526, 1646, 1649, 1685, 1691), гене gdh - группа v (положения 1886, 1937, 2028, 2282), гене pdhC - группа vi (положения 2513, 2775), гене pgm - группа vii (положение 3276).
IV. Проведение мультиплексной ПЦР
Мультиплексную амплификация генов abcZ, adk, aroE, fumC, gdh, pdhC, pgn N. gonorrhoeae, содержащих информативные однонунклеотидные полиморфизмы, проводят в две стадии:
На стадии (а) в качестве матрицы для амплификации используют препараты геномной ДНК N. gonorrhoeae, полученные на этапе (I), и первый набор специфичных праймеров. Таким образом, в ходе амплификации на стадии (а) происходит одновременная наработка 7 фрагментов генома N. gonorrhoeae. Реакционная смесь стадии (а) включает:
•1X ПЦР-буфер для HS Taq ДНК полимеразы (ЗАО «Евроген», Россия»);
• по 200 мкМ dATP, dCTP, dGTP, dUTP (ЗАО «Евроген», Россия»);
• смесь праймеров (концентрации приведены в Таблице 1) для стадии амплификации (а);
• 5 ед. HS Taq ДНК полимеразы (ЗАО «Евроген», Россия»);
• 1 мкл раствора ДНК геномной ДНК N. gonorrhoeae.
Мультиплексную ПЦР проводят на термоциклере S1000 (Bio-Rad, США). Для амплификации используют следующую программу:

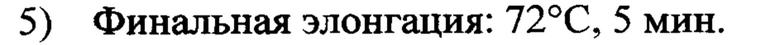
Далее проводят стадию амплификации (б), где в качестве матрицы используют ПЦР-продукты, полученные на стадии (а), и второй набор специфичных праймеров (Таблица 1), а также флуоресцентно меченый дезоксиуридинтрифосфат, позволяющий получать преимущественно одноцепочечные флуоресцентно-меченные ПЦР продукты. ПЦР-смесь стадии (б) включает:
• 1X ПЦР-буфер для HS Taq ДНК полимеразы (ЗАО «Евроген», Россия»);
• по 200 мкМ dATP, dCTP, dGTP, dUTP (ЗАО «Евроген», Россия»);
• 1 мкМ флуоресцентного субстрата Sulfo-Cyanine5 dUTP (ООО "Люмипроб РУС", Москва, Россия);
• смесь праймеров (концентрации приведены в Таблице 1) для стадии амплификации (б);
• 5 ед. HS Taq ДНК полимеразы (ЗАО «Евроген», Россия»); ж 1 мкл реакционной смеси, полученной на стадии (а).
Мультиплексную ПЦР проводят на термоциклере S1000 (Bio-Rad, США). Для амплификации используют следующую программу:

Реакционную смесь, полученную на стадии (б), используют для гибридизации на гидрогелевом биологическом микрочипе.
V. Проведение гибридизации и отмывки на биочипе
В отдельную пробирку добавляют 10 мкл буфера для гибридизации (0,3 М HEPES, рН 7,5, 3,0 М гуанидинтиоцианата, 30 мМ ЭДТА) и 20 мкл ПЦР-смеси. 30 мкл смеси вносят в гибридизационную камеру биологического микрочипа. Биологические микрочипы помещают в термостат и инкубируют при 37°С в течение 6-12 час. По окончании гибридизации биологические микрочипы трижды промывают дистиллированной водой и высушивают в потоке воздуха до полного исчезновения капель на поверхности подложки.
VI. Регистрация и интерпретация результатов гибридизации
Для регистрации гибридизационных картин используют Универсальный аппаратно-программный комплекс для анализа биологических микрочипов (УАПК) (ООО «БИОЧИП-ИМБ», Россия), оснащенный специализированным программным обеспечением «ImageWare».
Для каждого анализируемого образца геномной ДНК, выделенной из изолятов №1-3, получают соответствующую флуоресцентную картину гибридизации на биологическом микрочипе (Фигуры 2-4, соответственно).
Интерпретацию результатов гибридизации выполняют посредством сравнения интенсивностей флуоресцентного сигнала элементов, в которых образовались совершенные и несовершенные гибридизационные дуплексов. Сравнение флуоресцентных сигналов проводят внутри групп (i-vii) для каждого из 18 анализируемых положений. Максимальный сигнал элемента в группе соответствует выявленному однонуклеотидному полиморфизму. Так, на Фигуре 2 в группе i, соответствующей локусу abcZ, максимальные сигналы зарегистрированы в элементах '34 (С) 1' и '205 (А) 3'. Тем самым, исследуемый образец геномной ДНК изолята №1 включает информативные полиморфизмы С в положении 34 и А в положении 205 гена abcZ. Определение других информативных однонуклеотидных полиморфизмов проводят сходным образом, получая набор (последовательность) из 18 информативных полиморфизмов, составляющих нуклеотидный профиль. Полученный профиль сравнивают с эталонным из базы pubMLST, для которого известен генотип (Таблица 3), и получают искомый MLST генотип анализируемого изолята.
Таким образом, для первого изолята получают нуклеотидный профиль «CAGGGTCCGTTTTAGGTG» (Фигура 2), для второго изолята - нуклеотидный профиль «CAGGGGCCGTCCCAGATG» (Фигура 3), для третьего изолята «TGGGGTCCATTCCAGATC» (Фигура 4). Полученные нуклеотидные профили сравнивают по принципу максимального соответствия с эталонными профилями, соответствующими генотипам в базе pubMLST (Таблица 3), что позволяет определить генотип первого изолята как MLST 1594; второго - MLST 1901, третьего - MLST 9363. Корректность генотипирования с использованием предлагаемого способа проверяли методом секвенирования по Сэнгеру. Результаты, полученные с использованием способа, предлагаемого в данном изобретении, и полученные методом секвенирования, совпали для всех анализируемых образцов.
Предлагаемый в данном изобретении способ генотипирования изолятов N. gonorrhoeae посредством идентификации информативных однонуклеотидных полиморфизмов в генах «домашнего хозяйства» с помощью гибридизации на биологическом микрочипе, позволяет быстро и точно проводить процедуру генотипирования N. gonorrhoeae, существенно сокращая время и стоимость одного анализа. Метод не требует дорогостоящего оборудования и высококвалифицированного персонала. Результаты, полученные с использованием заявляемого способа, могут быть использованы для эпидемиологического мониторинга возбудителя гонококковой инфекции, контроля распространения мультирезистентных вариантов гонококка, в том числе, для выявления и характеризации изолятов N. gonorrhoeae, не являющихся эндемичными для отдельных географических регионов/стран, что может свидетельствовать о возможном трансграничном переносе таких изолятов.


--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="en"
nonEnglishFreeTextLanguageCode="ru" dtdVersion="V1_3"
fileName="Перечень последовательностей Кандинов ИД v.2.xml"
softwareName="WIPO Sequence" softwareVersion="2.3.0"
productionDate="2023-09-04">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>-</ApplicationNumberText>
<FilingDate></FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>-</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное учреждение науки Институт молекулярной биологии им. В.А.
Энгельгардта Российской академии наук (ИМБ РАН)</ApplicantName>
<ApplicantNameLatin>Engelhardt Institute of Molecular Biology,
Russian Academy of Sciences</ApplicantNameLatin>
<InventorName languageCode="ru">Кандинов Илья
Денисович</InventorName>
<InventorNameLatin>Kandinov I.D.</InventorNameLatin>
<InventionTitle languageCode="ru">СПОСОБ ГЕНОТИПИРОВАНИЯ ИЗОЛЯТОВ
NEISSERIA GONORRHOEAE НА БИОЛОГИЧЕСКОМ МИКРОЧИПЕ</InventionTitle>
<SequenceTotalQuantity>64</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>abcZ</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q131">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>forward primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aatcgtttatgtaccgcagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>abcZ</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q132">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>reverse primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagaacgagccgggatagga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q195">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>adk</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q196">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>forward primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aggcaggcacgcccttgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>adk</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q134">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>reverse primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caatacttcggctttcacgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>3</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>aroE</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q135">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>forward primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcggtcaatacgctggtg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>3</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>aroE</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q136">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>reverse primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgatgttgccgtacacata</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>fumC</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q137">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>forward primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tccggcttgccgtttgtcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>fumC</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q138">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>reverse primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttgtaggcggttttggcgac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>gdh</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q139">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>forward primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccttggcaaagaaagcctgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>gdh</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q140">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>reverse primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcgcacggattcattcgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>6</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>pdhC</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q141">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>forward primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctactacatcaccctgatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>6</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>pdhC</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q142">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>reverse primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atcggctttgatgccgtattt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>pgm</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q143">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>forward primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cggcgatgccgaccgcttgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>pgm</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q144">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>reverse primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtgatgatttcggttgcgcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>abcZ</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q145">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>forward primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aatcgtttatgtaccgcagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q32">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>abcZ</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q146">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>reverse primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccagtaacaatacatcgggc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>adk</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q147">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>forward primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtcaaagaacgcatcgcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q36">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>adk</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q148">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>reverse primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtaggtaacgtggtaagtacgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>17</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q38">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>3</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>aroE</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q149">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>forward primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>attgccaaccgtacccg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>17</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q40">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>3</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>aroE</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q150">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>reverse primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agagggcgtaggaagcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q42">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>fumC</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q151">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>forward primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctgaacaaaatcgccaacg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q44">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>fumC</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q152">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>reverse primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agttttcgttgaagctgttgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="23">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q48">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>gdh</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q153">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>forward primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccttggcaaagaaagcctgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="24">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q50">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>gdh</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q154">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>reverse primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gacagattcgatggtcagcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="25">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q52">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>6</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>pdhC</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q155">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>forward primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tatgccgaccgtatccgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="26">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q54">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>6</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>pdhC</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q156">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>reverse primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atcggctttgatgccgtattt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="27">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q56">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>pgm</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q157">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>forward primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aagtgttggacaacctgcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="28">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q58">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>pgm</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q158">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>reverse primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggaagcacgcatcagacc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="29">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q60">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>abcZ</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q159">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 34, SNP =
C</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggcgaaattcgcgatttatt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="30">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q62">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>abcZ</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q160">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 34, SNP =
T</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggcgaaatttgcgatttatt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="31">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q64">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>abcZ</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q161">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 205, SNP =
A</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gccggaaaacaaaaaaatcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="32">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q66">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>abcZ</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q162">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 205, SNP =
G</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gccggaaaacgaaaaaatcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="33">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q68">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>adk</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q163">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 565, SNP =
G</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtacattggcgcaagccgaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="34">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q70">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>adk</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q164">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 565, SNP =
A</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gacgcggtcgttgaaatcga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="35">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q72">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>adk</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q165">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 611, SNP =
G</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gacgcggtcgttgaaatcga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="36">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q74">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>adk</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q166">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 611, SNP =
A</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gacgcggtcattgaaatcga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="37">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q76">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>3</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>aroE</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q167">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 1112, SNP =
G</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggcggatgtgaacggcggtt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="38">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q78">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>3</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>aroE</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q168">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 1112, SNP =
A</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggcggatgtaaacggcggtt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="39">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q80">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>3</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>aroE</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q169">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 1271, SNP =
T</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgacaatcgggtgcgaaaaaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="40">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q82">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>3</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>aroE</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q170">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 1271, SNP =
G</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgacaatcgggggcgaaaaaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="41">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q84">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>fumC</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q171">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 1526, SNP =
C</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aatcaaaatccccgaaaacg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="42">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q86">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>fumC</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q172">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 1526, SNP =
G</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aatcaaaatgcccgaaaacg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="43">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q88">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>fumC</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q173">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 1646, SNP =
GC</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tatggcgggcgcgtcgggcaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="44">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q90">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>fumC</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q174">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 1646, SNP =
TA</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tatggcgggtgcatcgggcaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="45">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q92">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>fumC</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q175">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 1649, SNP =
TG</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tatggcgggtgcgtcgggcaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="46">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q94">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>fumC</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q176">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 1649, SNP =
CA</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tatggcgggcgcatcgggcaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="47">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q96">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>fumC</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q177">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 1685, SNP =
C</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tatgcccgtcatcgcctaca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="48">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q98">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>fumC</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>map</INSDQualifier_name>
<INSDQualifier_value>position = 1685, SNP =
T</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tatgcccgttatcgcctaca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="49">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q100">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>fumC</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q178">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 1691, SNP =
C</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccgttatcgcctacaacctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="50">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q102">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>fumC</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q179">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 1691, SNP =
T</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccgttatcgcttacaacctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="51">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q104">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>gdh</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q180">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 1886, SNP =
C</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>taaatacatcgaaagcgtgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="52">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q106">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>gdh</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q181">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 1886, SNP =
T</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>taaatacattgaaagcgtgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="53">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q108">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>gdh</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q182">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 1937, SNP =
C</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agagcgcggcgagttttacg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="54">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q110">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>gdh</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q183">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 1937, SNP =
T</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agagcgcggtgagttttacg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="55">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q112">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>gdh</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q184">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 2028, SNP =
A</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcccccgccagcttggatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="56">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q114">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>gdh</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q185">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 2028, SNP =
G</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcccccgccggcttggatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="57">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q116">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>gdh</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q186">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 2282, SNP
=G</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acgcatggcgggcaaagtgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="58">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q118">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>gdh</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q187">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 2282, SNP =
A</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acgcatggcaggcaaagtgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="59">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q120">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>6</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>pdhC</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q188">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 2513, SNP =
A</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtggaagcagacatctggt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="60">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q122">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>6</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>pdhC</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q189">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position =2513, SNP =
G</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtggaagcggacatctggt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="61">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q124">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>6</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>pdhC</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q190">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 2775, SNP =
T</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctgcgccgcttctttgaagt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="62">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q126">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>6</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>pdhC</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q191">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 2775, SNP =
C</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctgcgccgcctctttgaagt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="63">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q128">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>pgm</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q192">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 3276, SNP =
G</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gccaaagccgaatttgaagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="64">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q130">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>pgm</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q193">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>position = 3276, SNP =
C</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gccaaagcccaatttgaagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
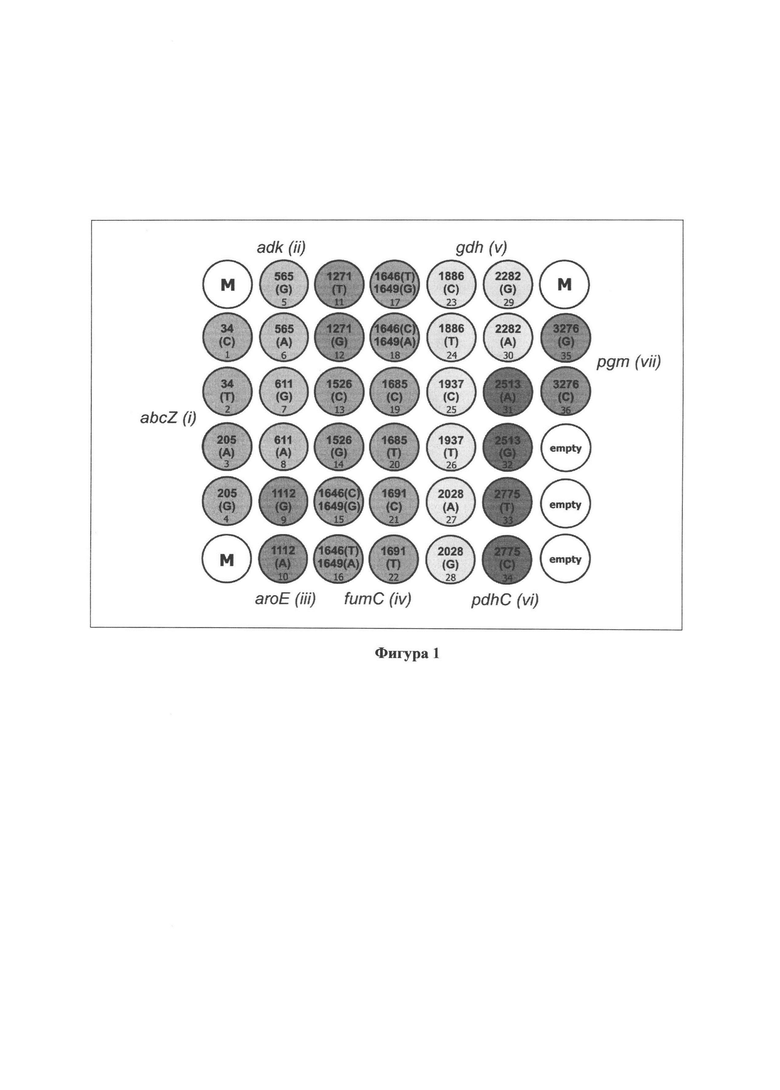



Изобретение относится к области молекулярной биологии. Описан способ генотипирования изолятов N. gonorrhoeae посредством идентификации информативных однонуклеотидных полиморфизмов в генах «домашнего хозяйства», с использованием биологического микрочипа. Техническим результатом изобретения является создание способа генотипирования изолятов N. gonorrhoeae посредством идентификации информативных однонуклеотидных полиморфизмов в генах «домашнего хозяйства» с помощью биологического микрочипа. 7 з.п. ф-лы, 4 ил., 4 табл., 1 пр.
1. Способ генотипирования изолятов N. gonorrhoeae посредством идентификации информативных однонуклеотидных полиморфизмов в генах «домашнего хозяйства», с использованием биологического микрочипа, включающий:
а) первый этап мультиплексной ПЦР при использовании геномной ДНК изолята N. gonorrhoeae в качестве матрицы и первого набора специфичных праймеров для амплификации фрагментов генов abcZ, adk, aroE, fumC, gdh, pdhC, pgm;
б) второй этап мультиплексной ПЦР, проводимой по асимметричному типу, с использованием в качестве матрицы ПЦР-продуктов, полученных на стадии (а), второго набора специфичных праймеров, а также смеси четырех дезоксинуклеозидтрифосфатов, в которой один из четырех дезоксинуклеозидтрифосфатов является флуоресцентно-меченым, что позволяет получать преимущественно одноцепочечные флуоресцентно-меченые фрагменты генов abcZ, adk, aroE, fumC, gdh, pdhC, pgm, последовательности которых содержат информативные однонуклеотидные полиморфизмы;
в) обеспечение биологического микрочипа для идентификации информативных однонуклеотидных полиморфизмов, представляющего собой подложку, содержащую множество дискретных элементов, в каждом из которых иммобилизован уникальный олигонуклеотидный зонд, имеющий последовательность, комплементарную последовательности одноцепочечного фрагмента, полученного на стадии (б) и выбранную из группы, включающей: i) последовательности фрагмента гена abcZ, содержащего информативные однонуклеотидные полиморфизмы в положениях 34 и 205; ii) последовательности фрагмента гена adk, содержащего информативные однонуклеотидные полиморфизмы в положениях 565 и 611; iii) последовательности фрагмента гена aroF, содержащего информативные однонуклеотидные полиморфизмы в положениях 1112 и 1271; iv) последовательности фрагмента гена fumC, содержащего информативные однонуклеотидные полиморфизмы в положениях 1526, 1646, 1649, 1685 и 1691; v) последовательности фрагмента гена gdh, содержащего информативные однонуклеотидные полиморфизмы в положениях 1886, 1937, 2028, 2282; vi) последовательности фрагмента гена pdhC, содержащего информативные однонуклеотидные полиморфизмы в положениях 2513, 2775; vii) последовательности фрагмента гена pdhC, содержащего информативные однонуклеотидные полиморфизмы в положении 3276;
г) гибридизацию амплифицированных флуоресцентно-меченых ПЦР-продуктов, полученных на стадии (б), на биологическом микрочипе, полученном на стадии (в), с образованием дуплексов с иммобилизованными зондами в условиях, обеспечивающих разрешение в один нуклеотид между образующимися в результате гибридизации совершенными и несовершенными дуплексами;
д) регистрацию и интерпретацию результатов гибридизации на биологическом микрочипе, проведенной на стадии (г), с использованием анализатора флуоресценции и программного обеспечения.
2. Способ по п. 1, характеризующийся тем, что на стадии (а) используют набор специфичных праймеров, последовательности которых представлены SEQ ID NO: 1-14.
3. Способ по п. 1, характеризующийся тем, что на стадии (б) используют набор специфичных праймеров, последовательности которых представлены SEQ ID NO: 15-28.
4. Способ по п. 1, характеризующийся тем, что на стадии (б) обратные праймеры используют, по меньшей мере, в десятикратном молярном избытке по отношению к прямым праймерам, что позволяет получать преимущественно одноцепочечные ПЦР продукты.
5. Способ по п. 1, характеризующийся тем, что на стадии (б) в качестве флуоресцентно-меченого дезоксинуклеозидтрифосфата используют флуоресцентно-меченый дезоксиуридинтрифосфат.
6. Способ по п. 1, характеризующийся тем, что биологический микрочип представляет собой подложку с гидрогелевыми элементами, полученными химическим способом или методом фотоиндуцируемой сополимеризации и содержащими иммобилизованные олигонуклеотидные зонды, последовательности которых представлены SEQ ID NO: 29-64.
7. Способ по п. 1, характеризующийся тем, что на стадии (д) регистрируют максимальный сигнал в каждой из групп, отвечающий образованию совершенного гибридизационного дуплекса, и устанавливают информативные однонуклеотидные полиморфизмы в каждом из анализируемых положений генов abcZ, adk, aroE, fumC, gdh, pdhC, pgm.
8. Способ по п. 1, характеризующийся тем, что на стадии (д) при интерпретации результатов получают уникальный нуклеотидный профиль, состоящий из последовательности информативных однонуклеотидных полиморфизмов, и устанавливают генотип изолята посредством сравнения полученного профиля с эталонными профилями из базы данных pubMLST.
| AU 2008240143 A1, 23.10.2008 | |||
| СПОСОБ МОЛЕКУЛЯРНОГО ТИПИРОВАНИЯ Neisseria gonorrhoeae | 2008 |
|
RU2380408C1 |
| Кубанов А.А., Соломка В.С., Шпилевая М.В., Вербенко Д.А., Дерябин Д.Г., Кандинов И.Д., Дементьева Е.И., Грядунов Д.А., Шаскольский Б.Л | |||
| Разнообразие NG-MAST- и MLST-сиквенс-типов у российских клинических изолятов Neisseria gonorrhoeae, несущих "мозаичную" аллель гена penA | |||
| Молекулярная | |||
Авторы
Даты
2024-04-04—Публикация
2023-09-28—Подача