Изобретение относится к области биотехнологии и генной инженерии растений, более конкретно к РНК-проводнику (молекуле, направляющей РНК, со специфичной нуклеотидной последовательностью) для контролирования экспрессии генов растений путем целенаправленного воздействия на нуклеотидную последовательность консервативного участка промоторной области гена PPD1 мягкой пшеницы системами редактирования генома CRISPR/Cas9, в которых молекулы РНК-проводника или сконструированный на его основе экспрессионный ДНК-вектор являются одним из компонентов.
Время колошения является одним из наиболее важных признаков мягкой пшеницы, варьирование которого позволяет адаптировать данную культуру к широкому спектру климатических условий. Одними из ключевых генов, позволяющих пшенице адаптироваться к регионам с различными эколого-климатическими условиями, являются PPD-1 (Photoperiod-1) гены, определяющие чувствительность/нечувствительность к фотопериоду, и VRN-1 гены, определяющие потребность растения в яровизации.
Генотипы пшеницы, нечувствительные в фотопериоду, несут доминантные гены, обозначенные индексом «a» (например, ген, расположенный на 2D хромосоме, обозначается как Ppd-D1a), данные генотипы пшеницы вызревают как в условиях длинного, так и короткого дня. Чувствительные к фотопериоду генотипы несут рецессивный ген, обозначенный индексом «b» (например, Ppd-D1b). Растения, чувствительные к фотопериоду, имеют задержку в сроках колошения на коротком дне (часто не успевают перейти к созреванию), в условиях длинного дня влияние этого гена на сроки колошения не изучалось.
Для генов Ppd-1a и Ppd-1b описано достаточно большое количество аллелей [1, 2, 3]. Но какой именно вклад в формирование фенотипа вносит каждый из них до сих пор плохо изучено. В работе (4) был изучен вклад различных нечувствительных к фотопериоду аллелей генов Ppd-1a в фотопериодический путь инициации колошения. Работа была проделана на материале интрогрессивных линий, и, таким образом, хотя в качестве реципиента был использован один и тот же сорт, исключать влияние дополнительных генетических локусов нельзя. Для понимания механизмов регуляции этих генов, необходимо анализировать мутации в условиях неизменной генетической среды. Большинство известных аллелей генов Ppd-1a мягкой пшеницы характеризуются обширными делециями в промоторной области от 1085 пн у Ppd-A1a.3 [3] до 2089 пн Ppd-D1a [1]. Интерес представляет более точное определение критичных для регуляции генов Ppd-1a областей и, возможно, идентификация сайтов связывания конкретных факторов, влияющих на транскрипцию.
Наиболее распространенным и сильным фактором раннего колошения мягкой пшеницы является нечувствительный к фотопериоду аллель Ppd-D1a, локализованный на 2D хромосоме и характеризующийся протяженной делецией 2 тпн в промоторной области [1, 5, 6]. Имеются данные, что аллель Ppd-D1a может ускорять время колошения на 3-5 дней даже в условиях длинного фотопериода [6, 7]. Аллель Ppd-D1a характеризуется повышенной экспрессией в ночной период, что положительно сказывается на времени перехода к колошению [4].
Значение времени от посева до колошения для производителей пшеницы высоко, поэтому селекционеры работают над сокращением вегетационного периода культурных сортов пшеницы. Ранее нами был описан способ получения линий яровой мягкой пшеницы с укороченным сроком колошения путем маркер-опосредованного отбора линий, содержащих аллели Ppd-D1a и Vrn-B3a [8]. Такой способ требует много времени и не исключает перенос нецелевых областей генома от сорта-донора. Возможность контролировать сроки колошения путем точечного изменения аллелей PPD1 позволит упростить процесс получения новых высокопродуктивных сортов, соответствующих современным требованиям. Получения новых аллелей генов PPD1, имеющих мутации в регуляторных областях гена на неизменном генетическом фоне, является технической проблемой, для решения которой необходимо расширение арсенала средств, доступных селекционерам. Настоящее изобретение решает эту проблему путем изменения последовательности промотора PPD1 средствами редактирования геномов растений CRISPR/Cas9.
Основными компонентами системы редактирования CRISPR/Cas9 являются белок Cas9, обладающий нуклеазной активностью, и РНК-проводник sgRNA, направляющая РНК специфичной композиции, которая направляет белок Cas9 к специфичному участку-мишени генома растений. РНК-проводник функционирует в качестве матрицы при редактировании ДНК и представляет собой химерную последовательность с элементами распознавания белка Cas9 и участка, комплементарного ДНК-мишени генома растения. Вследствие этого РНК-проводник состоит из двух областей - константной, длиной около 70 нуклеотидов, и вариабельной, длиной 17-25 нуклеотидов. Первая отвечает за образование функционального комплекса Cas9-sgRNA, а вариабельная - за специфичное связывания этого комплекса с целевой последовательностью ДНК в геноме [9]. Наиболее распространенная система использует PAM, состоящий из трех оснований NGG, он распознается нуклеазой Cas9 из Streptococcus pyogenes [10]. Разрезание целевой мишени возможно только при условии совпадения мишени и наличия проксимального участка 5′ РАМ-мотива, прилегающего к протоспейсеру.
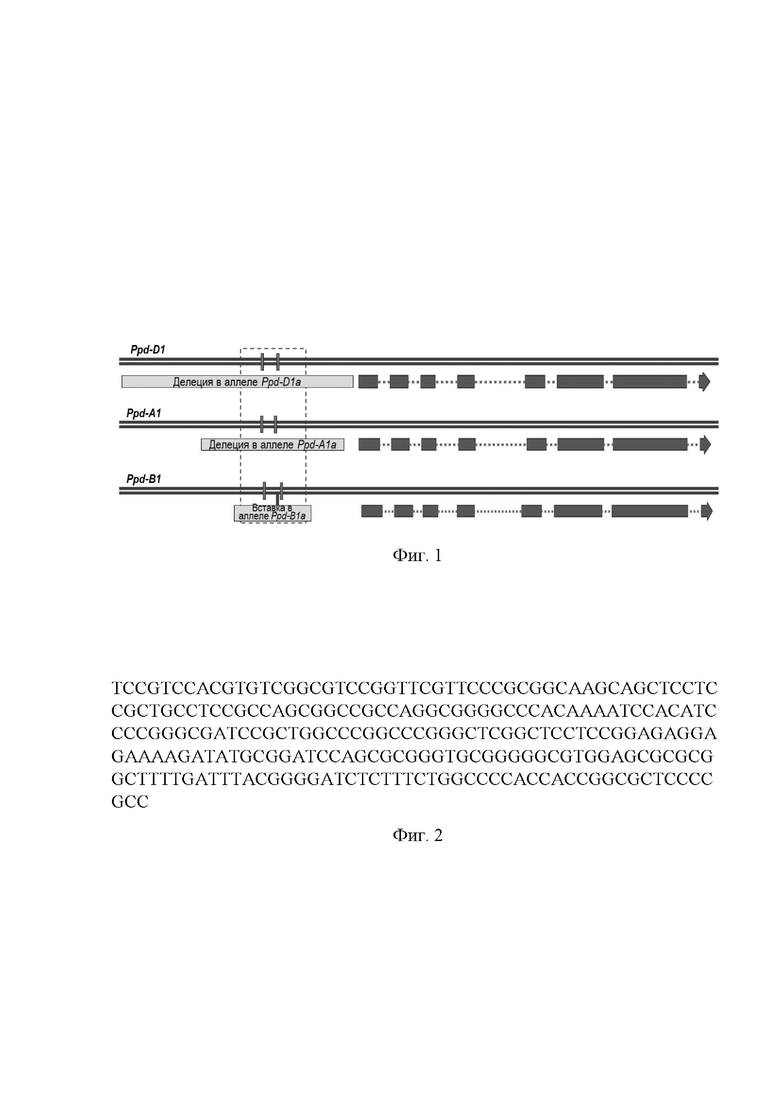
Задачей изобретения является разработка РНК-проводников, направляющих РНК со специфичной нуклеотидной последовательностью, которые могут быть использованы в системах CRISPR/Cas9 для внесения изменений в консервативный участок протяженностью 244 п.н. между - 707 и -951 п.н. промоторной области гена PPD-D1 (фиг. 1) без изменения других участков генома зерновых.
Задача решается, и технический результат достигается, путем создания молекулы РНК-проводника, где указанный РНК-проводник содержит первую нуклеотидную последовательность, выбранную из SEQ ID NO: 1 или SEQ ID NO: 2 и вторую нуклеотидную последовательность SEQ ID NO: 3, фланкирующую первую последовательность с 3'-конца и представляющую собой РНК-шпильку, при этом указанная первая последовательность обеспечивает связывание с целевым высококонсервативным участком промотора гена PPD-D1, а указанная вторая последовательность способна распознаваться РНК-направляемой ДНК-эндонуклеазой Cas9 с обеспечением внесения мутаций в целевой участок.
Принцип функционирования предлагаемых РНК-проводников базируется на особенностях работы системы CRISPR/Cas9. Нуклеаза Cas9 взаимодействует с представленными в настоящей заявке направляющими молекулами РНК-проводников, в результате чего образуется функциональный комплекс, который нацеливает белок Cas9 в специфическое местоположение внутри целевой последовательности промотора гена PPD-D1, комплементарной участку РНК-проводника. В результате такого взаимодействия функциональные домены комплекса CRISPR/Cas9 вносят разрывы в обе цепи ДНК, которые в дальнейшем восстанавливаются репарационными системами клетки. В отсутствии матрицы репарация проводится за счёт негомологичного соединения концов (NHEJ, non-homologous end joining). При этом в месте разрыва могут происходить вставки или делеции нуклеотидов, что вероятно приведет к образованию мутантных вариантов последовательности промотора гена PPD-D1.
В одном аспекте настоящее изобретение предусматривает возможность применения разрабатываемого РНК-проводника непосредственно в виде молекулы РНК и использование его в работах по геномному редактированию в составе рибонуклеопротеиновых комплексов (РНП), т.е. в системах CRISPR/Cas9 где комплекс Cas9-sgRNA может быть представлен в виде очищенного функционального белка Cas9 в смеси с молекулами РНК-проводника.
В другом аспекте настоящее изобретение предусматривает создание ДНК-векторов для интродукции в геном мягкой пшеницы последовательности разрабатываемого РНК-проводника с целью его стабильной экспрессии в отдельных клетках, тканях и растениях в результате генетической трансформации.
Примеры
Далее приводятся примеры, которые иллюстрируют возможные варианты осуществления изобретения.
Пример 1. Разработка РНК-проводника
Первый этап - подбор последовательностей-мишеней для внесения изменений в консервативный участок протяженностью 244 п.н. между -87 и -311 п.н. промоторной области гена PPD-D1 (фиг. 1) с помощью разрабатываемых РНК-проводников. Для этого амплифицировали фрагменты промоторной области гена PPD-D1 с геномной ДНК пшеницы линии Велют с использованием праймеров 5’-TGCGCCATGTAGGAATTG-3’ и 5’-GTGGTAGCTGAGATTGAGTATG-3’. ПЦР-продукты секвенировали по Сэнгеру. Последовательность участка PPD-D1 приведена на фиг. 2. Далее подбирали потенциальные мишени для РНК-проводников в целевой последовательности промотора гена PPD-D1. С этой целью использовали современные алгоритмы и программы, находящиеся в открытом доступе (11, 12). Основные критерии для выбора эффективных sgRNA были следующими: содержание GC от 30% до 80%, сохранение интактной вторичной структуры, отсутствие определенных мотивов в последовательности, отсутствие оффтергетных участков в геноме пшеницы. Поскольку выбор нуклеотидной последовательности РНК-проводника ограничен наличием РАМ последовательности (NGG) в целевой нуклеотидной последовательности промотора гена PPD-D1, авторы настоящего изобретения составили перечень из 10 потенциальных мишеней в участке между - 707 и -951 п.н. промоторной области рецессивного гена PPD-D1 (фиг. 3, фиг. 4). Матрицы для синтеза РНК-проводников в пробирке изготавливали с помощью олигонуклеотидов. Для этого к последовательности являющейся мишенью РНК-проводников без PAM сайта добавляли к 5'-концу последовательность нуклеотидов TTCTAATACGACTCACTATAG, а к 3'-концу - GTTTTAGAGCTAGAAATAGC. Получившиеся олигонуклеотиды смешивали с общим праймером AAAAGCACCGACTCGGTGCCACTTTTTCAAGTTGATAACGGACTAGCCTTATTTTAACTTGCTATTTCTAGCTCTAAAAC и достраивали на амплификаторе. Затем на созданных ДНК матрицах синтезировали РНК с помощью набора HiScribe T7 Quick High Yield RNA Synthesis Kit (New England Biolabs, США) по инструкции производителя.
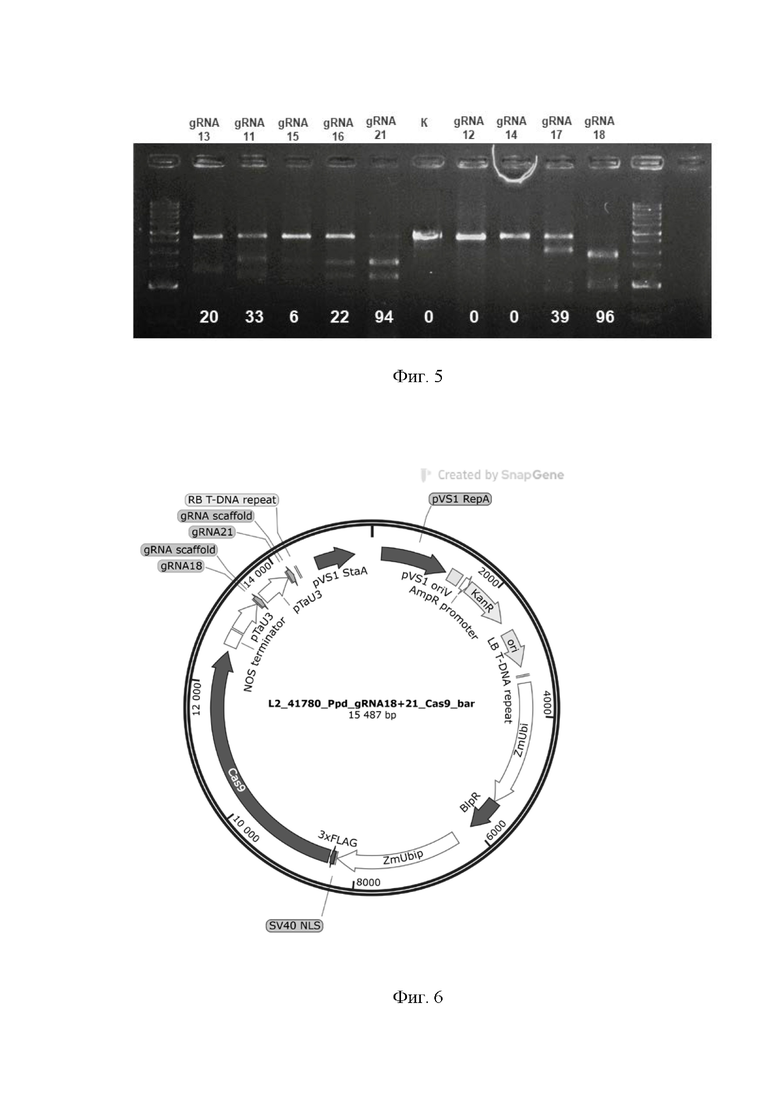
Далее проводили оценку эффективности РНК-проводников путем расщепления последовательности-мишени in vitro. На реакцию объемом 30 мкл брали молекулы РНК-проводника до концентрации 30 нМ, белок Cas9 (NEB) до концентрации 30 нМ и после совместной 10-минутной инкубации при комнатной температуре добавляли ПЦР-продукт гена-мишени до 3 нМ. В качестве контроля служили смеси ПЦР-продукта в той же концентрации без добавления Cas9 и РНК-проводника, или в присутствии Cas9, но без РНК-проводника. Инкубировали при 37°C в течение 120 мин, реакцию останавливали добавлением 1 мкл протеиназы K (30 мг/мл). Продукты реакции разделяли в 2% агарозном геле и фотографировали. Затем проводили денситометрический анализ для сравнительной оценки активности РНК-проводников. За 100% принимали интенсивность полосы нерасщепленного контроля; с этим значением сравнивали значения экспериментальных полос и определяли эффективность, вычитая полученное значение из 100 (фиг. 5). Анализ показал, что для внесения мутаций в целевые мишени в консервативном участке протяженностью 244 п.н. между - 707 и -951 промоторной области рецессивного гена PPD-D1 среди десяти изучаемых РНК-проводников эффективными являются РНК-проводники gRNA-18 (SEQ ID NO: 1), gRNA-21 (SEQ ID NO: 2). При их использовании, эффективность расщепления соответствующей целевой последовательности in vitro составила 96% и 94%, соответственно. Тогда как результативность комплексов Cas9 и РНК-проводников gRNA-11, gRNA-12, gRNA-13, gRNA-14, gRNA-15, gRNA-16 и gRNA-17 варьировала лишь в диапазоне 0-39%. Сравнительная оценка расщепляющей активности индивидуальных РНК-проводников показала, что для реализации технического результата изобретения предпочтительным вариантом является использование молекул РНК-проводников gRNA-18 (SEQ ID NO: 1) и gRNA-21 (SEQ ID NO: 2).
Пример 2. Разработка векторов для экспрессии в клетках однодольных зерновых культур
Одним из вариантов осуществления изобретения валяется применение РНК-проводников в составе экспрессирующих векторов. В данном примере последовательности трех наиболее эффективных РНК-проводников SEQ ID NO: 1 и SEQ ID NO: 2 использовали для разработки генетических конструкций, способных экспрессироваться в клетках однодольных зерновых культур. Указанные последовательности клонировали в вектор pFH85 (Addgene plasmid #128198; https://www.addgene.org/128198/; [13]), расщепленном по сайту рестрикции BpiI (Sibenzyme). После каждый из векторов, содержащий gRNA-18 или gRNA-21, вектор pFH33 (Addgene plasmid #128198; https://www.addgene.org/128198/; [13]), pICH47751 (Addgene plasmid #48002; https://www.addgene.org/48002/; [14]) для gRNA-18 и pICH47761 (Addgene plasmid #48003; https://www.addgene.org/48003/; [14]) для gRNA-21 расщепляли по сайту рестрикции BsaI (NEB) и лигировали для получения векторов L1-47751_prom_gRNA18_scaf и L1-47751_prom_gRNA21_scaf. Оба полученных таким образом вектора, а также вектора pFH66 (Addgene plasmid #131765; https://www.addgene.org/131765/; [13]) pFH114 (Addgene plasmid #128408; https://www.addgene.org/128408/; [13]) расщепляли по сайту рестрикции BpiI (Sibenzyme). Вектор pICSL4723-P1 (Addgene plasmid #86173; https://www.addgene.org/86173/; Youles SynBio plasmids) расщепляли по сайту рестрикции BsaI (NEB). Все эти расщепленные вектора лигировали вместе для получения векторной конструкции L2_41780_gRNA18+21_Cas9_bar (фиг. 6).
Исходя их этого, РНК-проводники для внесения мутаций в промоторную область гена PPD-D1 геном мягкой пшеницы содержат первую нуклеотидную последовательность, выбранную из SEQ ID NO: 1 или SEQ ID NO: 2, и вторую нуклеотидную последовательность SEQ ID NO: 3, фланкирующую первую последовательность с 3'-конца и, при этом указанная первая последовательность обеспечивает связывание с целевым высококонсервативным участком промотора гена PPD-D1, а указанная вторая последовательность способна распознаваться РНК-направляемой ДНК-эндонуклеазой Cas9 с обеспечением внесения мутаций в целевой участок. Плазмидный вектор, получивших название L2_41780_gRNA18+21_Cas9_bar (фиг. 6) обеспечивает возможность трансформации компетентных клеток E.coli с последующей наработкой большого количества копий. Очищенный препарат плазмидной ДНК указанного вектора может быть использован для биолистической агробактериальной трансформации мягкой пшеницы и содержит все необходимые компоненты, необходимые для последующего редактирования генома.
Пример 3. Геномное редактирование промоторной области гена PPD-D1 однодольных зерновых c помощью РНК-проводника на примере мягкой пшеницы линии Велют
Для получения растений с отредактированным геномом требуется взаимодействие комплексов Cas9-РНК-проводник с геномной ДНК. Полученную по примеру 2 конструкцию L2_41780_gRNA18+21_Cas9_bar, содержащую последовательности двух РНК-проводников gRNA-18 и gRNA-21, последовательность Cas9 и дополнительный селективный ген, переносили в клетки пшеницы с помощью генной пушки для внесения мутаций в промоторную область гена PPD-D1. В качестве эксплантов использовали эмбриогенные каллусы, инициированные из тканей незрелых зиготических зародышей мягкой пшеницы линии Велют (Triticum aestivum L.). В результате генетической трансформации отобрали 10 побегов-регенерантов. Наличие вставки последовательности РНК-проводника подтверждали ПЦР анализом тотальной ДНК, выделенной из растений-регенерантов. На фиг. 7 представлен образец агарозного геля после проведения ПЦР анализа. Подтвердили, что геном семи первичных предполагаемых трансформантов содержит последовательность РНК-проводников из вектора L2_41780_gRNA18+21_Cas9_bar (наблюдали амплификацию характерного фрагмента размером 682 п.н.), (фиг. 7).
Оценку эффективности РНК-проводника в способности обеспечить внесение мутаций в консервативную последовательность промотора PPD-D1 гена осуществляли через анализ трансгенных растений пшеницы V8, V11, V12, V15, V20, содержащих одновременно в геноме последовательности нуклеазы Cas9 и РНК-проводников. Для этого фрагмент гена PPD-D1 амплифицировали с помощью праймеров GCTTCTTCACACTACTCACTGTA/ CACACGGACTACTCCTCC. ПЦР-продукты выделяли из агарозного геля набором ZR Gel DNA Recovery Kit (Zymo Research) и секвенировали.
Анализ нуклеотидных последовательности показал, что пять из пяти независимых трансгенных растений, а именно растения V8, V11, V12, V15 и V20 содержали изменения нуклеотидной последовательности в сравнении с немодифицированной последовательностью рецессивного гена PPD-D1, присутствующего в геноме линии Велют (фиг. 8). Эти изменения возникли в результате функциональной активности РНК-проводников gRNA18 и gRNA 21, поскольку делеции обнаружены в ожидаемых сайтах расщепления молекулы ДНК комплексом Cas9-РНК-проводник (фиг. 8). Эффективность получения растений с мутантным вариантом консервативного участка промоторной области гена PPD-D1 составила 100% (5 из 5 растений-регенерантов, содержащих встройку вектора). Полученные растения содержали различные изменения, включающие единичную нуклеотидную делецию, единичную нуклеотидную вставку, делецию из трёх нуклеотидов; делецию из десяти нуклеотидов, делецию из 223 нуклеотидов, делецию из 266 нуклеотидов у трёх линий одновременно достигнуты и вставки, и делеции нуклеотидов (фиг. 8).
Использование заявляемого РНК-проводника, в составе экспрессионного вектора L2_41780_gRNA18+21_Cas9_bar в описываемом примере, позволило эффективно внести мутации в целевую нуклеотидную последовательность консервативного участка промоторной области гена PPD-D1 мягкой пшеницы.
Краткое описание чертежей
Фиг. 1. Нуклеотидные последовательности доминантных (PPD-1a) и рецессивных (PPD-1b) аллелей гена PPD1, несущие мутации в области промотора.
Фиг. 2. Последовательность фрагмента промоторной последовательности гена PPD-D1 (244 п.н.) пшеницы сорта Chinese spring использованная для разработки РНК-проводников.
Фиг. 3. Положение таргетных последовательностей на гене PPD-D1, являющихся целевыми мишенями для разработанных РНК-проводников в области расположения сайтов связывания транскрипционных факторов CHE в промоторной области.
Фиг. 4. Нуклеотидные последовательности мишеней для РНК-проводников указанных на фиг. 3.
Фиг. 5. Эффективность расщепления последовательностей-мишеней промоторной области гена PPD-D1. Представлена активность in vitro комплекса Cas9-РНК-проводник при разрезании целевого ДНК-субстрата. Визуализация реакции разрезания с помощью фрагментного анализа в 2% агарозном геле. В качестве отрицательного контроля (WT) использовали линеаризованный ДНК-субстрат мягкой пшеницы линии Велют в соответствующей концентрации, к которому не был добавлен комплекс эндонуклеазы и РНК-проводника. Интенсивность полосы нерасщепленного контроля принимали за 100% и сравнивали со значения экспериментальных полос денситометрически; эффективность расщепления (указана в нижней части геля) определяли как разность в % между WT и полученным значением для каждого отдельного РНК-проводника.
Фиг. 6. План-схема векторной конструкции L2_41780_gRNA18+21_Cas9_bar.
Фиг. 7. ПЦР анализ геномной ДНК независимых растений-регенерантов пшеницы, полученных в результате биобаллистического переноса разработанных РНК-проводников gRNA-18 и gRNA-21 в составе экспрессионного вектора L2_41780_gRNA18+21_Cas9_bar для внесения мутаций в промоторную область гена пшеницы PPD-D1. Наличие искомых фрагментов последовательности РНК-проводников размером 682 п.н. подтверждена у семи образцов. На фиг. 7 представлен агарозный гель, содержащий ПЦР-продукты РНК-проводников vr-31, где М - маркер молекулярного веса; K+ - положительный контроль (ДНК плазмиды L2_41780_gRNA18+21_Cas9_bar); К- - отрицательный контроль (нетрансгенное растение Велют), V1-V24 - отобранные независимые растения.
Фиг. 8. Генотипирование целевой нуклеотидной последовательности промоторной области PPD-D1 у полученных растений пшеницы методом секвенирования по Сэнгеру. Представлены нуклеотидные последовательности аллелей в целевом фрагменте контрольного растения (WT) и пяти независимых растений пшеницы V8, V11, V12, V15, V20 с изменениями (отклонениями в сравнении с WT), которые описаны справа от последовательности. Последовательности РНК-проводников gRNA-18 и gRNA-21 представлены сверху внутри стрелок. Пунктирной линией обозначен предпочтительный сайт разрезания ДНК.
Список литературы
1. Beales J., Turner A., Griffiths S. et al. (2007) A pseudo-response regulator is misexpressed in the photoperiod insensitive Ppd-D1a mutant of wheat (Triticum aestivum L.). Theor Appl Genet 115:721-33. https://doi.org/10.1007/s00122-007-0603-4
2. Wilhelm E.P., Turner A.S., Laurie D.A. (2009) Photoperiod insensitive Ppd-A1a mutations in tetraploid wheat (Triticum durum Desf.). Theor Appl Genet 118:285-294. https://doi.org/10.1007/s00122-008-0898-9
3. Nishida H., Yoshida T., Kawakami K. et al. (2013) Structural variation in the 5′ upstream region of photoperiod-insensitive alleles Ppd-A1a and Ppd-B1a identified in hexaploid wheat (Triticum aestivum L.), and their effect on heading time. Mol Breed 31:27-37. https://doi.org/10.1007/s11032-012-9765-0
4. Shaw L.M., Turner A.S., Laurie D.A. (2012) The impact of photoperiod insensitive Ppd-1a mutations on the photoperiod pathway across the three genomes of hexaploid wheat (Triticum aestivum). Plant J 71:71-84. https://doi.org/10.1111/j.1365-313X.2012.04971.x
5. Law C.N., Sutka J., Worland A.J. (1978) A Genetic study of day-length response in wheat. Heredity (Edinb) 41:185-191. https://doi.org/10.1038/hdy.1978.87
6. Worland A.J.J., Börner A., Korzun V. et al. (1998) The influence of photoperiod genes on the adaptability of European winter wheats. Euphytica 100:385-394. https://doi.org/10.1023/A:1018327700985
7. Киселева А.А., Егги Э.Э., Кошкин В.А. и др. (2014) Выявление генетических детерминант, определяющих различие почти изогенных линий Triticum aestivum L. по фотопериодической чувствительности. Генетика 50:802-813. https://doi.org/10.7868/S0016675814050075
8. Салина Е.А., Стасюк А.И., Киселёва А.А. Способ отбора растений яровой мягкой пшеницы с укороченным сроком колошения // Патент России RU 2710729 C1, 10.01.2020
9. Nishimasu H., Ran F.A., Hsu P.D. et al. (2014) Crystal Structure of Cas9 in Complex with Guide RNA and Target DNA. Cell 156:935-949. https://doi.org/10.1016/j.cell.2014.02.001
10. Hsu P.D., Lander E.S., Zhang F. (2014) Development and Applications of CRISPR-Cas9 for Genome Engineering. Cell 157:1262-1278. https://doi.org/10.1016/j.cell.2014.05.010
11. Doench J.G., Fusi N., Sullender M. et al. (2016) Optimized sgRNA design to maximize activity and minimize off-target effects of CRISPR-Cas9. Nat Biotechnol 34:184-191. https://doi.org/10.1038/nbt.3437
12. Cram D., Kulkarni M., Buchwaldt M. et al. (2019) WheatCRISPR: A web-based guide RNA design tool for CRISPR/Cas9-mediated genome editing in wheat. BMC Plant Biol 19:. https://doi.org/10.1186/s12870-019-2097-z
13. Hahn F., Korolev A., Sanjurjo Loures L., Nekrasov V. (2020) A modular cloning toolkit for genome editing in plants. BMC Plant Biol 20:. https://doi.org/10.1186/s12870-020-02388-2
14. Weber E., Engler C., Gruetzner R. et al. (2011) A Modular Cloning System for Standardized Assembly of Multigene Constructs. PLoS One 6:e16765. https://doi.org/10.1371/journal.pone.0016765
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="1.xml"
softwareName="WIPO Sequence" softwareVersion="2.3.0"
productionDate="2024-01-15">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1</ApplicationNumberText>
<FilingDate></FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>1</ApplicantFileReference>
<ApplicantName languageCode="ru">Киселёва Антонина
Андреевна</ApplicantName>
<ApplicantNameLatin>Antonina A. Kiseleva</ApplicantNameLatin>
<InventorName languageCode="ru">Киселёва Антонина
Андреевна</InventorName>
<InventorNameLatin>Antonina A. Kiseleva</InventorNameLatin>
<InventionTitle languageCode="ru">РНК-ПРОВОДНИКИ ДЛЯ ГЕНОМНОГО
РЕДАКТИРОВАНИЯ ПРОМОТОРНОЙ ОБЛАСТИ ГЕНА PPD-D1 МЯГКОЙ ПШЕНИЦЫ С
ПРИМЕНЕНИЕМ СИСТЕМЫ CRISPR/CAS9</InventionTitle>
<SequenceTotalQuantity>3</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q7">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggcggggagcgccggtggtg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tccgtccacgtgtcggcgtc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>76</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..76</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gttttagagctagaaatagcaagttaaaataaggctagtccgttatcaa
cttgaaaaagtggcaccgagtcggtgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| МОЛЕКУЛА РНК-ПРОВОДНИКА ДЛЯ ГЕНОМНОГО РЕДАКТИРОВАНИЯ ПРОТОМОТОРНОЙ ОБЛАСТИ ГЕНА VRN-A1 ОДНОДОЛЬНЫХ ЗЕРНОВЫХ С ПРИМЕНЕНИЕМ СИСТЕМЫ CRISPR/CAS9 | 2020 |
|
RU2762831C1 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ ПШЕНИЦЫ С НУКЛЕОТИДНОЙ ВСТАВКОЙ В ПРОМОТОРНОЙ ОБЛАСТИ ГЕНА VRN-A1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2020 |
|
RU2772578C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ ПШЕНИЦЫ С БИАЛЛЕЛЬНЫМИ МУТАЦИЯМИ В ПРОТОМОТОРНОЙ ОБЛАСТИ ГЕНА VRN-A1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2020 |
|
RU2772575C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ ПШЕНИЦЫ С НУКЛЕОТИДНОЙ ДЕЛЕЦИЕЙ В ПРОМОТОРНОЙ ОБЛАСТИ ГЕНА VRN-A1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2020 |
|
RU2772577C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С БИАЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2824558C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С ТРИАЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2817374C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С ТЕТРААЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2817383C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С НУКЛЕОТИДНОЙ ДЕЛЕЦИЕЙ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2817376C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С ТЕТРААЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2817384C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С ТРИАЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2817378C2 |


Изобретение относится к области биотехнологии, в частности к молекуле РНК-проводника sgRNA для внесения мутаций в консервативный участок промоторной области гена PPD-D1 мягкой пшеницы с применением системы редактирования генома CRISPR/Cas9. Изобретение позволяет эффективно получать РНК-проводники, направляющие РНК со специфичной нуклеотидной последовательностью, которые могут быть использованы в системах CRISPR/Cas9 для внесения изменений в консервативный участок промоторной области гена PPD-D1 без изменения других участков генома зерновых. 8 ил., 3 пр.
Молекула РНК-проводника sgRNA для внесения мутаций в консервативный участок промоторной области гена PPD-D1 мягкой пшеницы с применением системы редактирования генома CRISPR/Cas9, где молекула РНК-проводника sgRNA содержит первую нуклеотидную последовательность, выбранную из SEQ ID NO: 1, SEQ ID NO: 2 и вторую нуклеотидную последовательность SEQ ID NO: 3, фланкирующую первую последовательность с 3’-конца, при этом указанная первая последовательность обеспечивает связывание с целевым консервативным участком промотора гена PPD1, а указанная вторая последовательность способна распознаваться РНК-направляемой ДНК-эндонуклеазой Cas9 с обеспечением внесения мутаций в целевой участок.
| МОЛЕКУЛА РНК-ПРОВОДНИКА ДЛЯ ГЕНОМНОГО РЕДАКТИРОВАНИЯ ПРОТОМОТОРНОЙ ОБЛАСТИ ГЕНА VRN-A1 ОДНОДОЛЬНЫХ ЗЕРНОВЫХ С ПРИМЕНЕНИЕМ СИСТЕМЫ CRISPR/CAS9 | 2020 |
|
RU2762831C1 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ ПШЕНИЦЫ С БИАЛЛЕЛЬНЫМИ МУТАЦИЯМИ В ПРОТОМОТОРНОЙ ОБЛАСТИ ГЕНА VRN-A1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2020 |
|
RU2772575C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ ПШЕНИЦЫ С НУКЛЕОТИДНОЙ ВСТАВКОЙ В ПРОМОТОРНОЙ ОБЛАСТИ ГЕНА VRN-A1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2020 |
|
RU2772578C2 |
| ZAFAR, KASHAF et al | |||
| Employing template-directed CRISPR-based editing of the OsALS gene to create herbicide tolerance in Basmati rice, AoB PLANTS, 2023, vol | |||
| Прибор для нагревания перетягиваемых бандажей подвижного состава | 1917 |
|
SU15A1 |
| WO 2019120283 A1, 27.06.2019. | |||
Авторы
Даты
2024-07-04—Публикация
2024-01-25—Подача