Изобретение относится к области молекулярной биологии, а именно к способу генетической паспортизации сортов розы эфиромасличной с использованием ISSR-маркеров. Изобретение позволяет эффективно идентифицировать сорта розы эфиромасличной, составлять уникальные генетические формулы сортов и может быть использовано в селекции эфиромасличных культур.
Одним из главных приоритетов отечественного культивирования розы эфиромасличной является выведение сортов, близких по свойствам к болгарским сортам, являющимся мировым эталоном качества. Генетическая идентификация сортов занимает важное место среди селекционных исследований в розоводстве. Сведения о генотипе ценных сортов используются не только в научных целях, но и имеют практическое значение. В частности, на их основании составляются генетические паспорта сортов, используемые для селекции, а также для защиты прав селекционеров.
В настоящий момент пять сортов розы эфиромасличной коллекции ФГБУН «Научно-исследовательский институт сельского хозяйства Крыма» (Золушка, Лада, Лань, Легрина и Радуга) включены в «Государственный реестр селекционных достижений, допущенных к использованию» (далее - Госреестр). Нормативным документом для проведения сортоиспытания роз с целью внесения в Госреестр, в котором описаны все признаки, характерные для данного рода или вида, является «Методика проведения испытаний на отличимость, однородность и стабильность. Роза (Rosa L.)» Государственной комиссии по испытанию и охране селекционных достижений (RTG/0011/2 от 01.02.2007 г. № 12-06/02 https://gossortrf.ru/metodiki-ispytaniy-na-oos-2/). Однако современные научные достижения позволяют расширить перечень методов для идентификации сортов и сделать ее более точной. В связи с этим, генетический паспорт является надежным дополнением к результатам сортоиспытания по стандартным методикам, что подчеркивает актуальность проведения ДНК-паспортизации.
В основе создания генетического паспорта лежит анализ полиморфизма ДНК с помощью ДНК-маркеров. Наиболее распространенным методом анализа генетического разнообразия является полимеразная цепная реакция и основанные на ней аналитические подходы. Среди них популярен благодаря своей простоте и доступности анализ межмикросателлитных последовательностей - ISSR-анализ.
В связи с отсутствием способов паспортизации Rosa L. были изучены методики по другим культурам. Известен «Способ генетической паспортизации селекционных достижений малины на основе RAPD-маркеров» (Патент RU 2671678 C2; МПК C12Q 1/68; опубл. 06.11.2018 г., бюл. № 31). Недостатком данного способа является отсутствие конкретной формы самого генетического паспорта, поскольку даны сведения только по используемым RAPD-праймерам и полученным с их помощью спектрам, которые представлены на электрофореграмме.
Наиболее близким аналогом изобретения является методика изучения генотипов сортов сирени белорусской селекции с помощью RAPD- и ISSR-анализа (Спиридович Е.В., Молканова О.И., Коротков О.И., Власова А.Б., Юхимук А.Н., Решетников В.Н. Молекулярно-генетические методы в сохранении и изучении генофонда ботанических коллекций: материалы Международной конференции, посвященной 80-летию Центрального ботанического сада Национальной академии наук Беларуси, Минск, 19-22 июня 2012 г. - Минск: ГНУ «Центральный ботанический сад Национальной академии наук Беларуси», 2012. - С. 463-467). В этом способе проводится оценка генетического разнообразия сортов сирени и составлены генетические паспорта сортов на основании выявленных RAPD- и ISSR-маркеров по схеме: отбор образцов сирени; выделение ДНК из листьев, проведение RAPD- и ISSR-ПЦР; разделение продуктов ПЦР методом горизонтального электрофореза в агарозном геле; анализ результатов электрофореза с помощью программного обеспечения; кластеризация образцов с построением RAPD+ISSR дендрограммы, отражающей степень генетического сходства между сортами; составление генетических паспортов с указанием всех маркеров, полученных с помощью конкретных праймеров.
Однако, предлагаемый в известном способе набор праймеров предназначен для паспортизации сортов сирени и может быть неэффективным для использования при паспортизации сортов розы эфиромасличной. К недостаткам способа можно отнести использование RAPD-праймеров, имеющих такой существенный недостаток как низкая температура отжига, приводящая к образованию неспецифичных продуктов, которые могут быть ошибочно приняты за целевой фрагмент. При этом используется всего два ISSR-праймера, что не является достаточным для оценки генетического разнообразия и составления генетического паспорта. Также к недостаткам способа можно отнести использование при выделении ДНК меркаптоэтанола - токсичного вещества с неприятным запахом. Кроме того, в буфер для экстракции добавляется 1% поливинилпирролидона, однако для розы эфиромасличной предпочтительной является концентрация ПВП 2% в виду более высокого содержания загрязняющих веществ в ароматических растениях. Недостатком, хотя и не существенным, в структуре паспорта является отсутствие указания уникальных локусов, присутствующих только в конкретном сорте.
Задачей изобретения является разработка способа генетической паспортизации сортов розы эфиромасличной с использованием эффективных ISSR-праймеров, а также оптимизация протокола выделения ДНК для наиболее эффективной работы с материалом розы эфиромасличной.
Поставленная задача решается способом генетической паспортизации сортов розы эфиромасличной с использованием ISSR-маркеров, включающим отбор материала, выделение ДНК, ПЦР-амплификацию с отобранными праймерами, визуализацию результатов ПЦР методом горизонтального электрофореза, статистическую обработку полученных фрагментов, кластеризацию образцов с построением дендрограммы, отражающей их генетическую близость, а также составление генетического паспорта сорта, в котором согласно изобретению амплификацию ДНК проводят с последовательным участием восьми ISSR-праймеров, имеющих индивидуальную температуру отжига. При этом выделение ДНК проводят по модифицированному СТАВ-протоколу с использованием стеблей вместо листьев, двукратной промывкой смесью хлороформ/изоамиловый спирт и добавлением 2% поливинилпирролидона в СТАВ-буфер для экстракции. Генетический паспорт представляет собой совокупность генетических формул сорта, полученных при помощи используемых праймеров и содержащих уникальные полиморфные фрагменты, характерные только для данного сорта.
Преимущества способа определяются решением вышеописанной задачи изобретения.
В заявляемом способе используют исключительно ISSR-праймеры с индивидуально подобранной оптимальной температурой отжига. Увеличение количества применяемых ISSR-праймеров позволяет выявить большее количество уникальных полиморфных локусов, характерных только для данного сорта. Благодаря этому определяется уникальная ISSR-формула, описывающая данный сорт, на основании которой составляется генетический паспорт.
ДНК выделяют из стеблей, а не из листьев, поскольку в листьях эфиромасличных растений содержится большее количество эфирных масел, затрудняющих получение чистой ДНК. При выделении ДНК не используют токсичное вещество меркаптоэтанол, применяемое для очистки от белков, полифенолов и других органических ингибиторов, вместо этого применяют двукратную промывку смесью хлороформ/изоамиловый спирт для лучшей очистки от белков и липидов, а в СТАВ-буфер добавляют 2% ПВП, осаждающий полифенолы.
Способ осуществляют по следующей схеме:
1) Отбор образцов изучаемых сортов.
2) Выделение ДНК любым известным способом.
3) ПЦР-амплификация с восемью отобранными ISSR-праймерами (таблица 1) по известной методике.
4) Визуализация результатов ПЦР методом горизонтального электрофореза по стандартной методике.
5) Статистическая обработка полученных фрагментов с использованием известного программного обеспечения, кластеризация сортов общепринятым методом по генетической близости и выявление уникальных полиморфных локусов, на основании которых составляется генетический паспорт сорта, представляющий собой совокупность генетических формул сорта, полученных при помощи используемых праймеров и содержащих уникальные маркеры.
Пример осуществления способа
Отбор материала для исследований
Отбирали образцы 10 сортов розы эфиромасличной, произрастающих в полевых условиях: Аура, Белая, Искра, Казанлыкская, Свежен, Золушка, Лада, Лань, Легрина и Радуга (таблица 2).
Выделение ДНК
ДНК выделяли модифицированным СТАВ-методом: в качестве материала использовали не листья, а стебли, добавляли в стандартный СТАВ-буфер 2% поливинилпирролидона (ПВП) и проводили двукратную промывку смесью хлороформ/изоамиловый спирт.
100 мг ткани измельчали в ступке с 800 мкл СТАВ-буфера, переносили в пробирки и термостатировали при 65°С 1 час, перемешивая каждые 15 мин. Добавляли равное количество смеси хлороформ/изоамиловый спирт (24:1), смешивали интенсивным встряхиванием вручную до получения однородной суспензии и центрифугировали 10 мин при 14000g и 4°С. Отбирали верхнюю фазу в новые пробирки и повторяли предыдущий шаг. Переносили верхнюю фазу в новые пробирки, добавляли равный объем охлажденного при -20°С изопропанола и смешивали аккуратным переворачиванием. Оставляли на 30 мин при -20°С и центрифугировали при 1400 g и 4°С в течение 10 мин. Удаляли супернатант, промокали края и крышки пробирок салфеткой. Добавляли 70% этанол, охлажденный при -20°С, в объеме, равном объему изопропанола. Смешивали и центрифугировали 10 мин при 14000g и 4°С. Сливали супернатант, осаждали на вортексе и отбирали остатки спирта наконечником. Высушивали в термостате при 65°С с открытой крышкой в течение 5 мин. Добавляли 100 мкл буфера ТЕ, термостатировали при 65°С 10 мин и встряхивали на вортексе до растворения осадка. Полученный экстракт ДНК хранили при -20°С. Количество и качество выделенной ДНК определяли методом горизонтального электрофореза в 1,5% агарозном геле и на спектрофотометре SPECTROstar Nano («BMG LABTECH», Германия).
ПЦР-амплификация
ПЦР-смесь объемом 20 мкл имела следующий состав: 13,5 мкл деионизированной воды, 2 мкл 1-кратного буфера, 0,4 мкл дНТФ (10 ммоль), 2 мкл праймера (10 пмоль), 0,1 мкл Hot Start Taq ДНК-полимеразы (1000 единиц активности, «Qiagen», Германия) и 2 мкл ДНК (10 нг/мкл). Амплификация проводилась в термоциклере Т-100 («BIORAD», США по следующей программе (таблица 3).
Hot Start Taq
ДНК-полимеразы
Визуализация результатов ПЦР методом горизонтального электрофореза.
Разделение и визуализацию продуктов ПЦР проводили в 2%-агарозном геле с ТАЕ-буфером и бромистым этидием.
Статистическая обработка результатов, оценка генетической близости сортов и составление генетических паспортов.
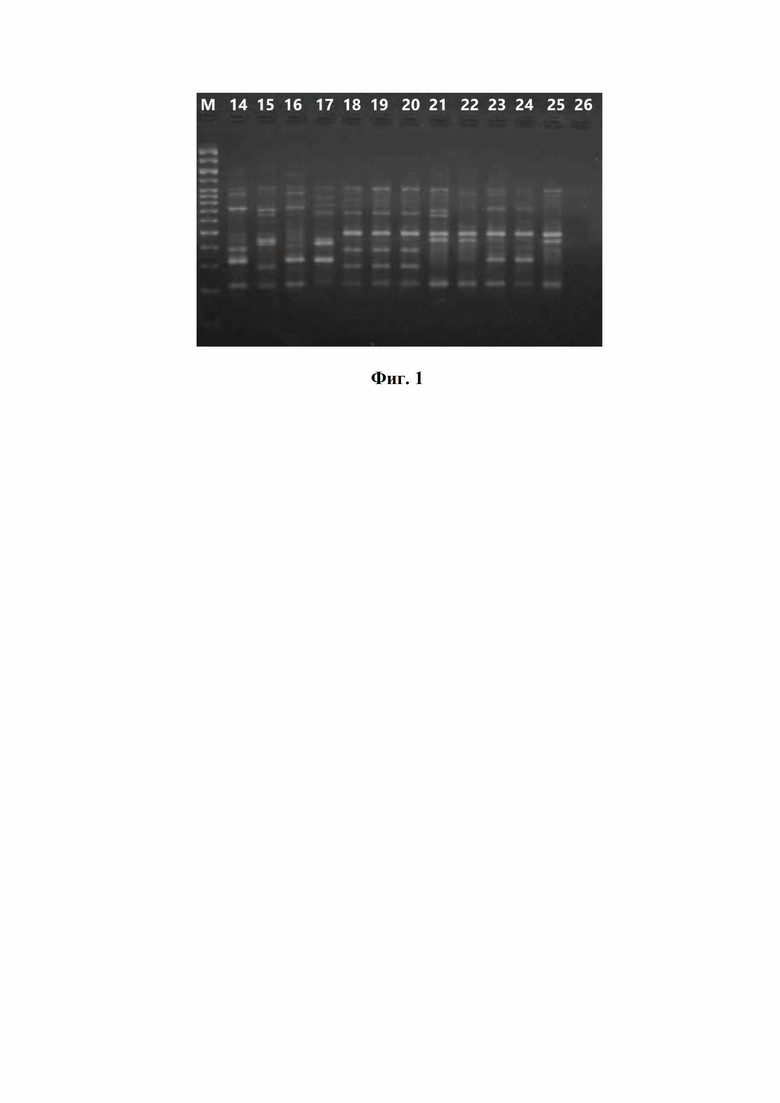
По результатам детекции продуктов ISSR-ПЦР присутствие фрагмента обозначали 1, а отсутствие - 0. На фиг 1. изображена электрофореграмма ISSR-спектров десяти сортов розы эфиромасличной, полученных при помощи одного из праймеров ISSR-840 (М - маркер длины ДНК, 14 - вид R. canina L., 15 - Аура, 16 - Белая, 17 - вид R. indica Major, 18 - Искра, 19 - Казанлыкская, 20 - Свежен, 21 - Золушка, 22 - Лада, 23 - Лань, 24 - Легрина, 25 - Радуга, 26 - отрицательный контроль). Виды R. canina L. и R. indica Major добавлены для иллюстрации генетических сходств при кластеризации).
На основании этих данных проводили кластеризацию образцов и оценивали генетическое родство отечественных сортов розы эфиромасличной к болгарским. Установлено, что сорт Золушка генетически близок к болгарским сортам.
Для исследуемых сортов розы эфиромасличной Золушка, Лань, Лада, Легрина и Радуга, включенных в «Государственный реестр селекционных достижений, допущенных к использованию», были составлены генетические формулы, представляющие собой все амплифицируемые фрагменты (локусы), выявленные при помощи каждого праймера, представленного в таблице 1.
В генетический паспорт включены только те формулы, которые содержат уникальные полиморфные фрагменты, характерные только для данного сорта.
В таблицах 4-8 приведены генетические паспорта исследуемых сортов розы эфиромасличной с указанием уникальных фрагментов (маркеров).
Преимуществом данной формы паспорта является возможность визуально оценить уникальные генетические характеристики сорта.
Таким образом, заявляемый способ позволяет проводить генетическую паспортизацию сортов розы эфиромасличной.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="primers.xml"
softwareName="WIPO Sequence" softwareVersion="2.3.0"
productionDate="2023-05-25">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2023110455</ApplicationNumberText>
<FilingDate>2023-04-24</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>1</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2023110455</ApplicationNumberText>
<FilingDate>2023-04-24</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное учреждение науки "Научно-исследовательский институт
сельского хозяйства Крыма"</ApplicantName>
<ApplicantNameLatin>Federalnoe gosudarstvennoe byudzhetnoe
uchrezhdenie nauki "Nauchno-issledovatelskii institut selskogo
khozyaistva Kryma"</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ генетической паспортизации
сортов розы эфиромасличной с использованием
ISSR-маркеров</InventionTitle>
<SequenceTotalQuantity>8</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>13</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q17">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agcagcagcagcy</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cagcagcagcagcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q19">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caacaacaacaacaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>17</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q21">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agagagagagagagagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>17</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agagagagagagagagt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>17</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagagagagagagagaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q23">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agagagagagagagagyt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagagagagagagagayt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБ ДНК-ИДЕНТИФИКАЦИИ И ГЕНЕТИЧЕСКОЙ ПАСПОРТИЗАЦИИ СОРТОВ РАЙГРАСА ПАСТБИЩНОГО И ОДНОЛЕТНЕГО НА ОСНОВЕ СИСТЕМ SSR- и SCoT-МАРКИРОВАНИЯ | 2023 |
|
RU2826148C1 |
| СПОСОБ МОЛЕКУЛЯРНО-ГЕНЕТИЧЕСКОЙ ИДЕНТИФИКАЦИИ СОРТОВ И ЛИНИЙ СОИ | 2024 |
|
RU2839697C1 |
| Способ подбора ДНК-маркеров для получения штаммспецифичных олигонуклеотидов к промышленным микроорганизмам и наборы штаммспецифичных олигонуклеотидов, разработанных этим способом для бактерий из видов Agrobacterium radiobacter и Paenibacillus polymyxa | 2023 |
|
RU2822667C1 |
| Способ мультиплексной идентификации 32 генетических маркеров льна | 2022 |
|
RU2804939C1 |
| Способ мультиплексной идентификации 52 генетических маркеров хозяйственно ценных признаков льна | 2023 |
|
RU2826718C1 |
| НАБОР ОЛИГОНУКЛЕОТИДОВ И СПОСОБ ОБОГАЩЕНИЯ ГЕНОМНОЙ КДНК ВИРУСА КРЫМСКОЙ-КОНГО ГЕМОРРАГИЧЕСКОЙ ЛИХОРАДКИ ГЕНОТИПА ЕВРОПА-1 (V) МЕТОДОМ ПОЛИМЕРАЗНОЙ ЦЕПНОЙ РЕАКЦИИ | 2023 |
|
RU2839313C1 |
| СПОСОБ ГЕНОТИПИРОВАНИЯ ИЗОЛЯТОВ NEISSERIA GONORRHOEAE НА БИОЛОГИЧЕСКОМ МИКРОЧИПЕ | 2023 |
|
RU2816767C1 |
| Способ сполиготипирования микобактерий туберкулезного комплекса с использованием ДНК-амплификации в иммобилизованной фазе и биологический микрочип для его осуществления | 2023 |
|
RU2807998C1 |
| СПОСОБ ОПРЕДЕЛЕНИЯ СУБЛИНИЙ ВОЗБУДИТЕЛЯ ТУБЕРКУЛЕЗА ЛИНИИ L2 Beijing НА БИОЛОГИЧЕСКИХ МИКРОЧИПАХ | 2022 |
|
RU2790296C1 |
| Нуклеотидная последовательность, кодирующая фермент литиказу, и панель олигонуклеотидов для получения синтетической нуклеотидной последовательности гена литиказы | 2023 |
|
RU2826150C1 |
Изобретение относится к биотехнологии, а именно к способу генетической паспортизации сортов розы эфиромасличной с использованием ISSR-маркеров. Изобретение позволяет эффективно идентифицировать сорта розы эфиромасличной, составлять уникальные генетические формулы сортов и может быть использовано в селекции эфиромасличных культур. 3 з.п. ф-лы, 1 ил., 8 табл., 3 пр.
1. Способ генетической паспортизации сортов розы эфиромасличной с использованием ISSR-праймеров, включающий отбор материала, выделение ДНК, ПЦР-амплификацию с отобранными праймерами, визуализацию результатов ПЦР методом горизонтального электрофореза, статистическую обработку полученных фрагментов, кластеризацию образцов с построением дендрограммы, отражающей их генетическую близость, составление генетического паспорта сорта, отличающийся тем, что амплификацию ДНК проводят с последовательным участием следующих ISSR-праймеров:
2. Способ по п. 1, отличающийся тем, что используют ISSR-праймеры с индивидуально подобранной оптимальной температурой отжига (°C):
ISSR-А34 52,0
ISSR-В10 52,4
ISSR-В13 41,9
ISSR-230/41 58,0
ISSR-807 52,6
ISSR-812 52,4
ISSR-834 56,2
ISSR-840 48,9.
3. Способ по п. 1, отличающийся тем, что выделение ДНК проводят по модифицированному СТАВ-протоколу с использованием стеблей вместо листьев, двукратной промывкой смесью хлороформ/изоамиловый спирт и добавлением 2% поливинилпирролидона в СТАВ-буфер для экстракции.
4. Способ по п. 1, отличающийся тем, что генетический паспорт представляет собой совокупность генетических формул сорта, полученных при помощи используемых праймеров и содержащих уникальные полиморфные фрагменты, характерные только для данного сорта.
| БОРОННИКОВА С.В | |||
| Генетическая паспортизация популяций редких видов растений рода Adonis с использованием ISSR- и IRAP-маркеров // Известия ТСХА | |||
| Колосоуборка | 1923 |
|
SU2009A1 |
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| КОСТЮКОВА E | |||
| E., и др | |||
| Молекулярно-генетический анализ редких видов орхидных Брянской области // Разнообразие растительного мира | |||
| Многоступенчатая активно-реактивная турбина | 1924 |
|
SU2013A1 |
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| СУХАРЕВА А.С | |||
| и др | |||
| ДНК-маркеры для генетического | |||
Авторы
Даты
2023-09-05—Публикация
2023-04-24—Подача