Настоящее изобретение относится к области молекулярной биологии и биотехнологии и описывает способ подбора штамм-специфичных ДНК-маркеров промышленных штаммов микроорганизмов для конструирования комплементарных олигонуклеотидов, применяемых для детекции интродуцированных промышленно ценных микроорганизмов после применения. Также представляются наборы штаммспецифичных олигонуклеотидов, разработанных данным способом для бактерий из видов Agrobacterium radiobacter и Paenibacillus polymyxa.
Использование биопрепаратов на основе живых культур микроорганизмов широко распространено в сельскохозяйственной практике. Они обладают различными полезными свойствами для растений: ростстимуляция, азотфиксация, фосфатмобилизация, защита от патогенов и другие. Молекулярная идентификация штаммов бактерий, составляющих основу биопрепаратов, стала обязательным элементом для паспортизации и патентного депонирования коллекционных и промышленных микроорганизмов. Она также выявляет генетический полиморфизм изучаемых штаммов по сравнению с другими микроорганизмами данного вида.
Одним из решений по подбору ДНК-маркеров для выявления бактерий в различных образцах является способ по патенту RU 2329305 (опубл. 20.07.2008, конвенционный приоритет 01.03.2001 US 60/272,642), предусматривающий амплификацию ДНК-матрицы в клиническом образце посредством ПЦР-РВ, где используют праймеры и не менее двух флуорогенных зондов. При этом каждый праймер комплементарен сегменту гена 16S рРНК, консервативному для разнообразных видов эубактерий. Праймеры ограничивают область гена 16S рРНК, содержащую консервативную и дивергентную части. Однако, решения, предлагаемые этим патентом, направлены на широкую группу эубактерий и не позволяют идентифицировать отдельные штаммы.
Известен способ генетической детерминации штаммов Bacillus thuringiensis для их паспортизации и определения биовара с помощью мультиплексной ПЦР и последующего пиросеквенирования (патент RU 2627178, опубл. 03.08.2017). Однако по данному методу невозможно детектировать отдельный штамм из-за его не строгой специфичности.
В патенте RU 2376388 (опубл. 20.12.2009) представлен набор синтетических олигонуклеотидов для выявления ДНК возбудителя болезней овощных культур - грамотрицательной бактерии Pseudomonas corrugata методом полимеразной цепной реакции, который позволяет точно идентифицировать вид патогенных бактерий P. corrugata, в свою очередь недостатком метода является отсутствие штаммспецифичности.
Изобретение по патенту RU 2395583 (опубл. 27.07.2010) относится к способу и набору, с помощью которых можно избирательно детектировать живые клетки микроорганизма, присутствующие в продуктах питания или клинических образцах. К неоспоримым плюсам данного способа относится возможность дифференцировать клетки в различных объектах, отличая живые и не живые микроорганизмы. Однако недостатком метода является невозможность определить штамм микроорганизма, а в указанных в патенте примерах маркеры подбираются на консервативных участках генов, отвечающих за патогенность бактерий.
Способ по патенту RU 2233888 (опубл. 10.08.2004) используется для определения в любом исследуемом материале (кровь, тканевая жидкость и другие биологические материалы больных людей и животных) ДНК риккетсиеподобного микроорганизма «Montezuma». Недостатком является отсутствие специфических праймеров и модификаций основной методики для определения нового вида микроорганизма, а также необходимость постановки двух ПЦР реакций - сначала на внешней паре праймеров, а затем с использованием гнездовых праймеров.
Работа RU 2209831 (опубл. 10.08.2003) направлена на решение проблемы с ложноположительными пробами на ген дифтерийного токсина, который содержится в Corynebacterium diphtheriae. Разработанный способ позволяет функционально дифференцировать микроорганизмы, обладающие активным геном в рамках одного вида. Недостатком методики является слабая специфичность, направленная скорее на группу штаммов.
В литературе также встречается информация о видо- или штамм-специфичных маркерах и праймерах к ним на диагностически важные микроорганизмы (Sato N. et al., 2014; Silva K.G. et al., 2022; Hernández I. et al., 2020). В описанных случаях для поиска маркеров авторами были использованы последовательности ДНК, кодирующие информацию о белках, с небольшими различиями в методах выявления SNPs (однонуклеотидный полиморфизм - участок для подбора маркеров). При этом полногеномное секвенирование до сих пор достаточно дорогостоящая процедура и требующая тщательной биоинформатической обработки данных.
Ближайшим аналогом может быть принят способ диагностики биоматериалов на наличие в них агробактерий, описанный в патенте RU 2458142 (опубл. 10.08.2012), где детекцию фитопатогенных бактерий Agrobacterium в различных биоматериалах осуществляли с помощью ПЦР в реальном времени (ПЦР-РВ). Однако минусом данного способа является невозможность детекции определенного штамма Agrobacterium и то, что авторы создали диагностический комплекс, способный детектировать только патогенные агробактерии.
Все указанные методы были ориентированы на детекцию патогенов человека, животных и растений, с чем связано отсутствие у них штаммспецифичности в конечном итоге. Современное развитие науки позволяет с помощью метагеномного анализа выявлять весь пул микроорганизмов в конкретном исследуемом субстрате. Однако это не гарантирует выявления заданных промышленно ценных или клинически важных штаммов. При этом будут значительные трудозатраты и увеличенная себестоимость.
Целью настоящего изобретения является разработка способа подбора уникальных маркерных участков ДНК, способных детектировать интродуцированные промышленно ценные микроорганизмы в местах их применения (растения, почва и другие) и имеющих свойство генетического паспорта. Также целью изобретения является конструирование разработанным способом наборов штаммспецифичных олигонуклеотидов для бактерий Agrobacterium radiobacter 204 и Paenibacillus polymyxa П13 НК.
Техническим результатом будет маркерная область ДНК, представленная SNPs или другими нуклеотидными заменами (делеции и/или инсерции) и сконструированные комплементарные олигонуклеотидные праймеры. Маркерная область будет являться для выбранного штамма его генетическим паспортом. Заявляемый способ предполагает удешевление процедуры паспортизации бактерий и получения результатов по детекции интродуцированных штаммов при сохранении высокого уровня точности. Кроме того, результатом изобретения будут являться наборы штаммспецифичных олигонуклеотидов для бактерий Agrobacterium radiobacter 204 и Paenibacillus polymyxa П13 НК.
Указанная задача решена способом подбора ДНК-маркеров для получения штаммспецифичных олигонуклеотидов к промышленным микроорганизмам, включающим выделение ДНК из чистой культуры микроорганизмов, определение нуклеотидной последовательности межгенного региона целевого штамма с помощью секвенирования по методу Сэнгера, подбор ближайших гомологов для полученной последовательности с охватом концевых участков генов и межгенного региона и проведение их множественного выравнивания, выявление в выровненных последовательностях штаммспецифичных участков ДНК и конструирование на их основе олигонуклеотидов для проведения ПЦР-РВ.
Также цель достигается конструированием наборов штаммспецифичных олигонуклеотидов, которые включают праймеры с последовательностями SEQ ID NO: 2 и 3 - для детекции штамма Agrobacterium radiobacter 204 и SEQ ID NO: 5 и 6 - для детекции штамма Paenibacillus polymyxa П13 НК.
В настоящем изобретении предложен способ подбора молекулярного маркера согласно известным нуклеотидным последовательностям межгенных регионов хромосомной ДНК искомых и референтных штаммов. Это позволяет точно определить штаммспецифичный участок ДНК и сконструировать на его основе олигонуклеотиды, комплементарные только к данному штамму. Этот подход способствует быстрому достижению технического результата и минимизации финансовых затрат.
Как описано выше для детекции микроорганизмов в большинстве изобретений используются олигонуклеотиды, сконструированные на основе нуклеотидных последовательностей, кодирующих белки. Предлагаемый способ основан на высокой вариабельности межгенных участков хромосом бактерий, где возможно выявление штаммспецифичных ДНК-маркеров. Возможно также подобрать такие маркеры, которые будут отсутствовать не только у ближайших родственников и гомологов, но и у отдаленных таксонов живых организмов. Последнее критически важно при исследовании таких богатых видовым и штаммовым разнообразием сред обитания микроорганизмов как почва и ризоплана растений.
Способ осуществляют по следующей схеме:
1. Выделение ДНК из чистой культуры микроорганизмов любым известным способом.
2. Определение нуклеотидной последовательности межгенного региона целевого штамма с помощью секвенирования по методу Сэнгера.
3. Подбор ближайших гомологов для полученной в п. 2 последовательности с охватом концевых участков генов и межгенного региона в базе данных ДНК и проведение их множественного выравнивания друг с другом.
4. Выявление в выровненных последовательностях штаммспецифичных участков ДНК (ДНК-маркеров: инсерций / делеций / замен) и конструирование на их основе олигонуклеотидов для проведения ПЦР в реальном времени (ПЦР-РВ).
Примеры осуществления способа.
Следующие ниже примеры представлены исключительно для целей иллюстрации реализации данного изобретения и никоим образом не должны рассматриваться как ограничивающие объем настоящего изобретения.
В качестве целевых микроорганизмов использовались штаммы Agrobacterium radiobacter 204 и Paenibacillus polymyxa П13НК из Крымской коллекции микроорганизмов ФГБУН «НИИСХ Крыма» (https://ckp-rf.ru/usu/507484/?sphrase_id=9090231), являющиеся живой биологической основой Комплексного биопрепарата (патент RU 2777194, 01.08.2022), разработанного для оптимизации симбиотрофного питания, контроля фитопатогенов и увеличения продуктивности растений (далее - биопрепарат).
Краткое описание графических изображений.
На фиг. 1 представлена часть ITS-региона штамма A. radiobacter 204 при выравнивании с последовательностями из ГенБанка и праймер 204SpPr3F, подобранный к штаммспецифичному участку; в позициях 21, 24, 26, 28 и 36 отмечены полиморфизмы у праймера и ближайших гомологов.
На фиг. 2 представлена часть ITS-региона штамма A. radiobacter 204 при выравнивании с последовательностями из ГенБанка и праймер 204SpPr3R, подобранный к штаммспецифичному участку; в позициях 595-600, 603, 610-613 отмечены полиморфизмы у праймера и ближайших гомологов, в позиции 596 - уникальный полиморфизм (SNP, single nucleotide polymorphism).
На фиг. 3 представлена часть YeGyS-региона штамма P. polymyxa П13НК при выравнивании с последовательностями из ГенБанка и праймер PanPr8F, подобранный к штаммспецифичному участку; в позициях 355, 358, 367 отмечены полиморфизмы у праймера и ближайших гомологов, в позиции 362 - SNP.
На фиг. 4 представлена часть YeGyS-региона штамма P. polymyxa П13НК при выравнивании с последовательностями из ГенБанка и праймер PanPr8R, подобранный к штаммспецифичному участку; в позиции 758 отмечен полиморфизм у праймера и ближайших гомологов.
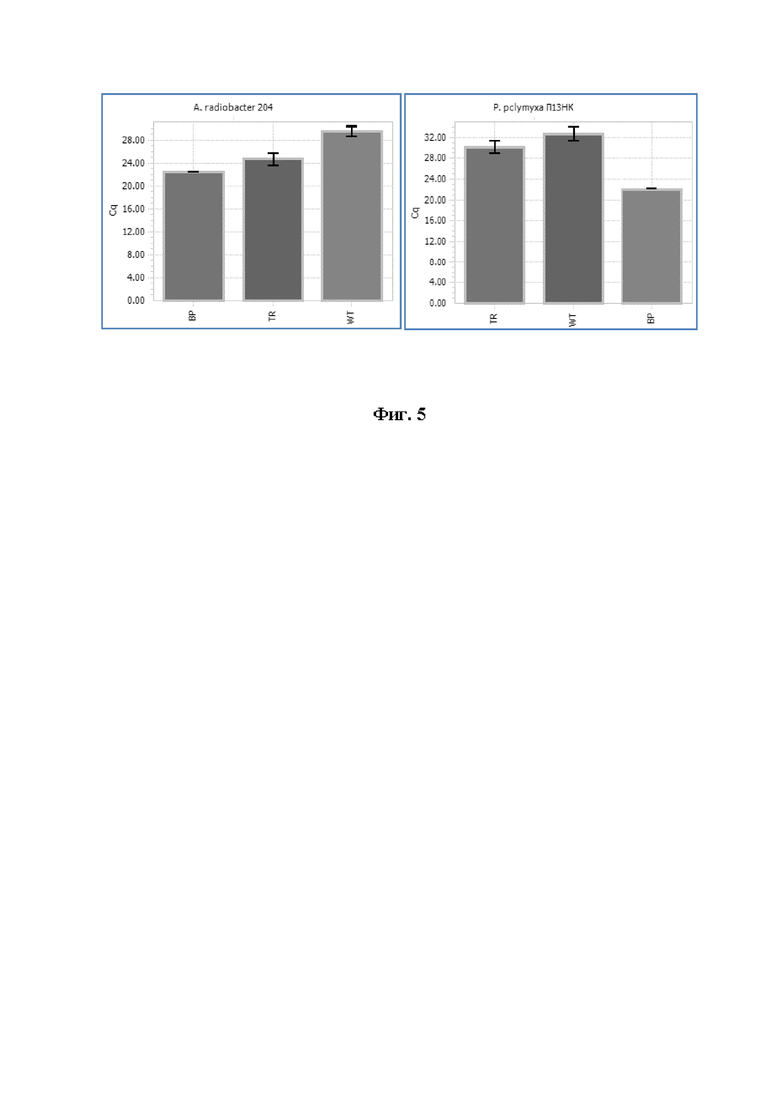
На фиг. 5 представлен анализ приживаемости коллекционных штаммов в ризоплане Sorghum bicolor (5 суток после прорастания). После обработки семян биопрепаратом с помощью олигонуклеотидов живые агенты биопрепарата A. radiobacter 204 и P. polymyxa П13НК обнаружены на корнях 5-суточных проростков, о чем свидетельствует меньший цикл ПЦР с штаммспецифичными праймерами.
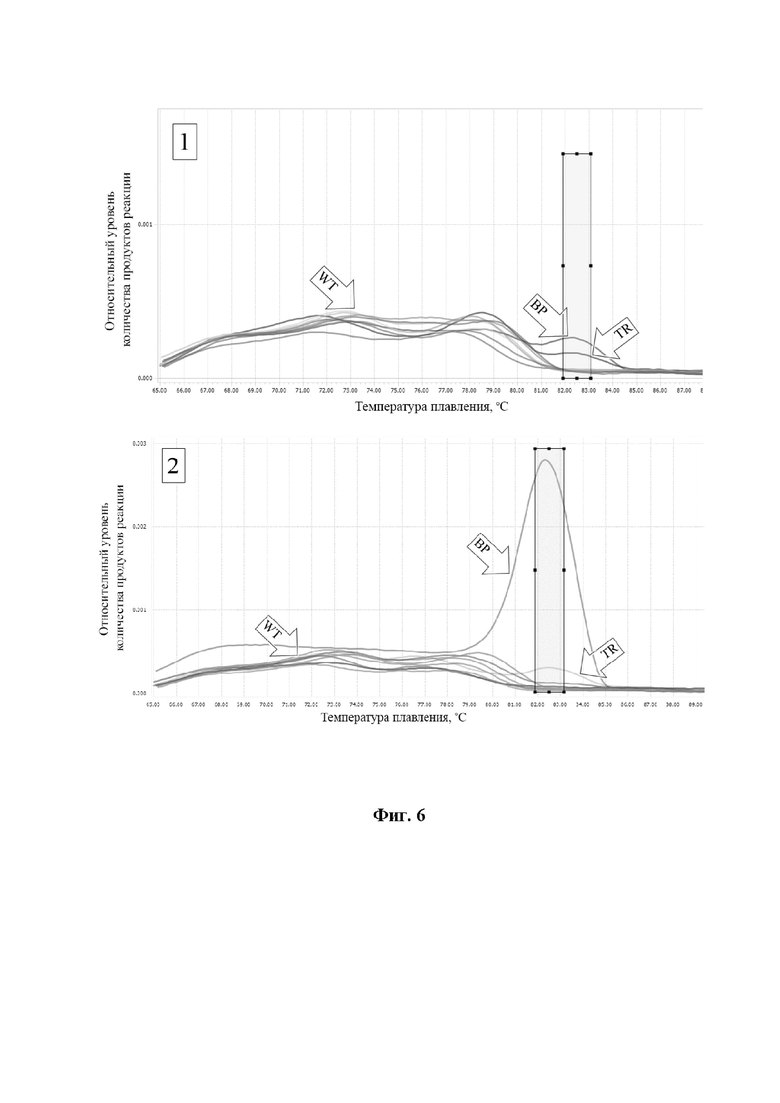
На фиг. 6 представлен анализ приживаемости P. polymyxa П13НК из биопрепарата в ризосфере S. bicolor (14 суток после прорастания). Выявление P. polymyxa П13НК в ризосфере сорго зернового при применении биопрепарата в традиционной системе земледелия (1) и системе no-till (2).
Пример 1. Целевой микроорганизм Agrobacterium radiobacter 204, маркерная область ДНК: ITS.
На первом этапе работы для определения нуклеотидной последовательности ITS (internal transcribed spacer, внутренний транскрибируемый спейсер) штамма A. radiobacter 204 использована чистая культура бактерий. Ее ДНК выделяли коммерческим набором GeneJet genomic DNA purification kit (ThermoFisher, США) согласно протоколу производителя для грамотрицательных бактерий.
Далее проводили реакцию ПЦР и секвенирования с праймерами FGPS 1490-72 (5'-tgcggctggatcccctcctt-3') и FGPL 132-28 (5'-ccgggtttccccattcgg-3'), описанными С. Риффардом с соавторами (Riffard S. et al., 1998). Реакцию ПЦР проводили в смеси общим объемом 20 мкл со следующим составом: 1х буфер для полимеразы, 0,5 U Taq-ДНК полимераза (Qiagen, Germany), 1,5 мМ Mg2+, 1 ммоль прямого и обратного праймеров (Евроген, Россия), 0,2 ммоль каждого из четырех dNTP (Евроген, Россия) и ≈20 нг ДНК-матрицы. Реакцию проводили в амплификаторе Т100 Thermal Cycler (Bio-Rad, США) по следующей программе: 3 мин при 95°C (начальная денатурация); далее 35 циклов - 30 с при 95°C (денатурация), 30 с при 45-65°C (отжиг), 1 мин при 72°C (элонгация); 10 мин при 72°C (заключительная элонгация). Продукты амплификации были очищены от ПЦР-смеси с помощью набора CleanUp Standart (Евроген, Россия) по протоколу производителя. Очищенные ПЦР-продукты использовали как матрицу для реакции секвенирования по методу Сэнгера, которую осуществила компания Евроген (Россия) на генетическом анализаторе GeneticAnalyzer 3500xL (Applied Biosystems, США) согласно протоколу производителя. Для большей точности ампликоны проанализированы с двух сторон как по плюс цепи ДНК, так и по комплементарной ей. Получили последовательность SEQ ID NO: 1.
На следующем этапе к последовательности SEQ ID NO: 1 подобрали 18 ближайших гомологичных последовательностей в ГенБанке NCBI. Подбор осуществляли на общедоступном ресурсе nucleotide BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi?PROGRAM=blastn&PAGE_TYPE=BlastSearch&LINK_LOC=blasthome), опираясь на следующие параметры работы: систематическое положение, близкое к нашему целевому микроорганизму (вид, род), максимальная идентичность (Percent identity) и покрытие (Query Cover), минимальное математическое ожидание (Expected value). Множественное выравнивание последовательностей проводили в программе BioEdit (Hall, 1999) по алгоритму ClustalW со стандартными настройками.
На четвертом этапе «вручную» выбирали целевые маркерные участки с основным требованием: наличие участков цепи ДНК, отличающихся в какой-либо позиции от подобранных гомологичных последовательностей. Штаммспецифичная область для штамма A. radiobacter 204 на 5'-конце определена в позиции 21-54 нуклеотидных оснований (фиг. 1), а на 3'-конце позициями 588-628 (фиг. 2). Ориентируясь на эти участки, были сконструированы штаммспецифичные олигонуклеотиды (праймеры) для использования в ПЦР-РВ. С помощью общедоступного ресурса PrimerBLAST (https://www.ncbi.nlm.nih.gov/tools/primer-blast/) подбирали праймеры со следующими параметрами: длина - 20-22 нуклеотида, 3'-конец должен захватывать маркерный участок, температура комплементарного отжига на ДНК-матрице должна составлять не менее 60°С, отсутствие у праймеров гомологии в Генбанке NCBI, особенно среди бактерий и микроскопических грибов, которые могут обитать в почве. Таким образом подобраны праймеры: 204SpPr3F (SEQ ID NO: 2) и 204SpPr3R (SEQ ID NO: 3) (фиг. 1 и 2). Характеристика новых праймеров: длина по 20 нуклеотидов, максимально возможная температура отжига 62,5°С, длина продукта 571 п.н., отсутствуют полные гомологи в базе данных.
Пример 2. Целевой микроорганизм Paenibacillus polymyxa П13НК, маркерная область ДНК: YeGyS (yheC/yheD-gyraseB spacer) - некодирующая область, расположенная между уникальным для P. polymyxa геном синтеза оболочки спор и геном β-субъединицы ДНК-гиразы
На первом этапе работы из чистой культуры бактерий была выделена ДНК штамма P. polymyxa П13НК. Выделение провели коммерческим набором GeneJet genomic DNA purification kit (ThermoFisher, США) согласно протоколу производителя для грамположительных бактерий.
Далее проводили реакцию ПЦР и секвенирования на YeGyS-регион с праймерами PSpPr2F (5'-cgtcaccaatcatcacagcg-3') и PSpPr2R (5'- ccagagagccgtacaatgct-3'), разработанными авторами специально для заявляемого изобретения. Для проведения ПЦР и секвенирования использованы реактивы и параметры, аналогичные Примеру 1. YeGyS для штамма P. polymyxa П13НК секвенирован и последовательность представлена под номером SEQ ID NO: 4.
На следующем этапе к последовательности SEQ ID NO: 4 подобрали 9 ближайших гомологичных последовательностей в ГенБанке NCBI. Подбор осуществляли на общедоступном ресурсе nucleotide BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi?PROGRAM=blastn&PAGE_TYPE=BlastSearch&LINK_LOC=blasthome), опираясь на параметры работы, указанные в Примере 1. Множественное выравнивание последовательностей проводили в программе BioEdit (Hall, 1999) по алгоритму ClustalW со стандартными настройками.
На четвертом этапе «вручную» выбирали целевые маркерные участки в YeGyS спейсере ДНК штамма P. polymyxa П13НК с наличием отличающихся в какой-либо позиции от подобранных гомологичных последовательностей: для прямого праймера этот регион был в позициях 350-380 п.н. (фиг. 3), для обратного - 737-780 (фиг. 4). Ориентируясь на эти участки, с помощью PrimerBLAST были сконструированы штаммспецифичные олигонуклеотиды (праймеры) для использования в ПЦР-РВ со следующими параметрами: длина - 20-22 нуклеотида, 3ʹ-конец должен захватывать маркерный участок, температура комплементарного отжига на ДНК-матрице должна составлять не менее 60°С, отсутствие у праймеров гомологии в Генбанке NCBI, особенно среди бактерий и микроскопических грибов, которые могут обитать в почве. Таким образом подобраны праймеры: PanPr8F (SEQ ID NO: 5) и PanPr8R (SEQ ID NO: 6) (фиг. 3 и 4). Характеристика новых праймеров: длина 20 и 22 нуклеотида соответственно, максимально возможная температура отжига 64°С, длина продукта 395 п.н., отсутствуют полные гомологи в базе данных.
Оценку функциональности выявленных ДНК-маркеров и созданных на их основе наборов олигонуклеотидов для штаммов Agrobacterium radiobacter 204 и Paenibacillus polymyxa П13 НК проводили в условиях лабораторного и полевого экспериментов.
Постановка лабораторного эксперимента. Семена Sorghum bicolor стерилизовали 10 % раствором пероксида водорода в течение 5 мин, затем обильно промывали стерильной дистиллированной водой. Раскладывали в предварительно простерилизованные системы для проращивания семян, состоящих из чашек Петри и фильтровальной бумаги. Семена перед посевом обработали биопрепаратом и проращивали в стерильных условиях в течение 5 суток. В контрольном варианте семена обработали стерильной водой.
Выделение ДНК. По прошествии указанного срока для детекции штамма в ризоплане сорго из нее выделяли суммарную ДНК. Выделение осуществляли с помощью набора PowerSoil DNA Isolation Kit (Qiagen, Германия) по протоколу производителя. Для гомогенизации образцов использовали VortexGenee-2 (Mo-Bio, США).
Постановка ПЦР. Выделенную ДНК использовали как матрицу (10 нг на реакцию) при проведении ПЦР-РВ. В реакции использовался набор БиоМастер HS-qPCR SYBR Blue (2×) (Биолабмикс, Россия) согласно протоколу производителя и штаммспецифичные праймеры из расчета 5 пмоль/реакцию каждого. Отрицательным контролем была ДНК, выделенная из ризопланы контрольного варианта без инокуляции, положительным - чистый биопрепарат. Режим проведения ПЦР соответствовал используемой в наборе HS-полимеразы и экспериментально подобранной температуре отжига праймеров.
Анализ результатов. Согласно полученному результату (фиг. 5) показано, что выход продуктов реакции в варианте с обработкой (вариант TR) наступает раньше для A. radiobacter 204 на 5 циклов, а для P. polymyxa П13НК на 2 цикла, чем без обработки биопрепаратом (WT). Выход продуктов реакции положительного контроля (ВР) составил 22 цикла. Ранний выход продуктов реакции свидетельствует о стабильной работе праймеров и косвенно о количестве бактерий в образце. Кроме того, анализ кривых плавления опытного образца показал полную идентичность с положительным контролем и значительное отличие контроля без обработки.
Постановка полевого эксперимента. Эксперимент проводили на опытном поле ФГБУН НИИСХ Крыма в с. Клепинино, почва чернозем южный. Семена Sorghum bicolor перед посевом обработали биопрепаратом и высевали в сроки, рекомендованные для данного региона (первая декада мая), в контрольном варианте семена обработали водой. Растения выращивали по двум технологиям: традиционная с применением вспашки и no-till без пахоты. После 2 недель вегетации с корней сорго была отобрана ризосфера.
Выделение ДНК. По прошествии указанного срока для детекции штамма в ризосферной почве из нее выделяли суммарную ДНК. Выделение осуществляли с помощью набора PowerSoil DNA Isolation Kit (Qiagen, Германия) по протоколу производителя. Для гомогенизации образцов использовали VortexGenee-2 (Mo-Bio, США).
Постановка ПЦР. Выделенную ДНК использовали как матрицу (10 нг на реакцию) при проведении ПЦР-РВ. В реакции использовался набор БиоМастер HS-qPCR SYBR Blue (2×) (Биолабмикс, Россия) согласно протоколу производителя и штаммспецифичные праймеры из расчета 5 пМоль/реакцию каждого. Отрицательным контролем была ДНК, выделенная из ризосферы контрольного варианта без инокуляции, положительным - чистый биопрепарат. Режим проведения ПЦР соответствовал используемой в наборе HS-полимеразы и экспериментально подобранной температуре отжига праймеров.
Анализ результатов. Согласно полученному результату (фиг. 6) показано, что температура плавления продуктов ПЦР-РВ в варианте с обработкой биопрепаратом (вариант TR) для штамма P. polymyxa П13НК совпадает с положительным контролем (ВР) и составляет 82,4°С.
Представленные результаты свидетельствуют о функциональности заявляемого способа подбора ДНК-маркеров и способности разработанных на его основе комплементарных олигонуклеотидов качественно детектировать интродуцированные промышленно ценные микроорганизмы в местах их применения. Дополнительным положительным аспектом является возможность быстрого мониторинга приживаемости промышленных микроорганизмов после применения и учет времени их действия. Также возможно выявление нелицензионного их использования.
На основании изложенного показано, что представленные примеры подтверждают достижение заявленного технического результата и промышленную применимость заявленного изобретения.
--->
<?xml version="1.0" encoding="ISO-8859-1"?>
<!DOCTYPE ST26SequenceListing SYSTEM "ST26SequenceListing_V1_3.dtd"
PUBLIC "-//WIPO//DTD Sequence Listing 1.3//EN">
<ST26SequenceListing productionDate="2023-12-13"
softwareVersion="2.3.0" softwareName="WIPO Sequence"
fileName="днк-маркер перечень последов3.xml" dtdVersion="V1_3">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText/>
<FilingDate/>
</ApplicationIdentification>
<ApplicantFileReference>1</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное учреждение науки "Научно-исследовательский институт
сельского хозяйства Крыма"</ApplicantName>
<ApplicantNameLatin>FSBSI Research Institute of Agriculture of
Crimea</ApplicantNameLatin>
<InventorName languageCode="ru">Абдурашитов Сулейман
Февзиевич</InventorName>
<InventorNameLatin>Abdurashytov Suleiman</InventorNameLatin>
<InventionTitle languageCode="ru">Способ подбора ДНК-маркеров для
получения штаммспецифичных олигонуклеотидов к промышленным
микроорганизмам и наборы штаммспецифичных олигонуклеотидов,
разработанных этим способом для бактерий из видов Agrobacterium
radiobacter и Paenibacillus рolуmуха</InventionTitle>
<SequenceTotalQuantity>6</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>643</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..643</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Agrobacterium radiobacter</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgccggaccgacccggcgaccggactatcgtcctgtatgggtggttgatcga
agactttgttttgattggcttaccggtgcacagagatgggcccgtagctcagttggttagagcacacgct
tgataagcgtggggtcggaagttcaagtcttcccgggcccaccattggtttgctgaatttacctgatccc
tggcggtttgactgtcgggtgtttgcgatggttggggctgtagctcagctgggagagcacctgctttgca
agcagggggtcagcggttcgatcccgctcagctccaccaatcgcggaacgcggtttgggtgtgtagcgcg
ggaatatccttctgaagaaataaaagtttgcatcgttcgcaaggactgatgcctgttctgaatacattgt
gaagagaagatatgtctggaagcttccaggtgttttaagcccttgtggtttgaagcgtccgagcccagtc
ccagagaaaccatgtgatggcttactcggccggaattggcggagggattggaggtaggtaggaaagcttg
tcctgggcatgttttgttgtttgaaggtttacgccttcatctgatggacatggctggatttgatgttgcc
tgaccgcgcatcaccggatgatatctcgaag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cggactatcgtcctgtatgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtccatcagatgaaggcgta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>785</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..785</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Paenibacillus polymyxa</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctcgaaatcagcaaagcccgttacccacaaggtcattatcatgacagatgcg
gatgtcgatggtgcccatattcgtacgttgttgctgacgttcttctatcggtacatgcgtaaaatcattg
atgcaggctatatttatattgctcagccaccactattcaaagtggagcgtaacaaagttgtacgttatgc
gaactccgaggcggaacgtgatgagattatcagggaatttggagaaaacgcgaaatacaatgtacagcgc
tataaaggcttgggtgagatgaatgcgacccaactttgggaaactacgatggaccctgagagtcgtacta
tgcttcaggtaactgtcagtgacgcgatgttggctgatacactgttcaataccttgatgggtgataatgt
agaacctcgtcgtgactttatccaagaacatgcgaagtacgtgaaaaatcttgatttttaatgtgtgatg
ttccttcctagtggctgtattacgtgctcattaggtagtgttttgtaagagttcacaaggggcgcccaaa
tgggcgtcccttttcttctcattaaccagcccgggtgtatttgaatatttgaactcattgttttaggctt
ttttcttagagcgtatggttttacccgtggatgaagagtaccggccgtagcctactgcatatctgaatat
tttatgatagaggcttttgtttggaagggaagagaacaccttaattgctggtctcgtattaacctccaga
atccacgggataattttgctggtccaatcccac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgcgatgttggctgatacac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggaggttaatacgagaccagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Набор олигонуклеотидных праймеров и флуоресцентно-меченых зондов и способ выявления ДНК возбудителей сапа и мелиоидоза методом ПЦР с детекцией продукта в режиме реального времени | 2019 |
|
RU2738358C1 |
| СПОСОБ ИДЕНТИФИКАЦИИ И ДИФФЕРЕНЦИАЦИИ ПРОКАРИОТИЧЕСКИХ ОРГАНИЗМОВ | 2011 |
|
RU2486251C2 |
| Олигонуклеотиды для определения мутации S:N501Y SARS-CoV-2 | 2022 |
|
RU2791958C1 |
| Олигонуклеотиды для определения 12FAB серотипа Streptococcus pneumoniae | 2021 |
|
RU2795021C1 |
| Олигонуклеотиды для определения 22FA серотипа Streptococcus pneumoniae | 2021 |
|
RU2802203C2 |
| Олигонуклеотиды для определения 8 серотипа Streptococcus pneumoniae | 2021 |
|
RU2802081C2 |
| Набор олигонуклеотидных праймеров Ft 182 и способ определения бактерий Francisella tularensis (Варианты) | 2019 |
|
RU2706564C1 |
| Набор олигонуклеотидных праймеров Ft 101 и способ определения бактерий Francisella tularensis (варианты) | 2019 |
|
RU2703803C1 |
| Комплексный биопрепарат для оптимизации минерального питания растений, защиты от фитопатогенов, повышения продуктивности и способ получения этого биопрепарата | 2022 |
|
RU2777194C1 |
| Набор олигонуклеотидных праймеров Ft 40 и способ определения бактерий Francisella tularensis (Варианты) | 2019 |
|
RU2706570C1 |




Изобретение относится к области молекулярной биологии. Описан способ подбора ДНК-маркеров для получения штаммспецифичных олигонуклеотидов к промышленным микроорганизмам. Также описаны наборы штаммспецифичных олигонуклеотидов, полученных заявленным способом для детекции штамма Agrobacterium radiobacter 204 и штамма Paenibacillus рolуmуха П13НК. Технический результат заключается в получении результатов по детекции интродуцированных штаммов при сохранении высокого уровня точности. 3 н.п. ф-лы, 6 ил., 2 пр.
1. Способ подбора ДНК-маркеров для получения штаммспецифичных олигонуклеотидов к промышленным микроорганизмам, включающий выделение ДНК из чистой культуры микроорганизмов, определение нуклеотидной последовательности межгенного региона целевого штамма с помощью секвенирования по методу Сэнгера, подбор ближайших гомологов для полученной последовательности с охватом концевых участков генов и межгенного региона и проведение их множественного выравнивания, выявление в выровненных последовательностях штаммспецифичных участков ДНК и конструирование на их основе олигонуклеотидов для проведения ПЦР-РВ.
2. Набор штаммспецифичных олигонуклеотидов, полученных способом по п.1, для детекции штамма Agrobacterium radiobacter 204, который включает праймеры с последовательностями SEQ ID NO: 2 и 3.
3. Набор штаммспецифичных олигонуклеотидов, полученных способом по п.1, для детекции штамма Paenibacillus рolуmуха П13НК, который включает праймеры с последовательностями SEQ ID NO: 5 и 6.
| Способ генетической паспортизации штаммов Bacillus thuringiensis с помощью проведения мультиплексного экспресс-анализа референтных последовательностей ДНК | 2015 |
|
RU2627178C2 |
| Cai, P., Han, M., Zhang, R | |||
| et al | |||
| SynBioStrainFinder: A microbial strain database of manually curated CRISPR/Cas genetic manipulation system information for biomanufacturing | |||
| Выбрасывающий ячеистый аппарат для рядовых сеялок | 1922 |
|
SU21A1 |
Авторы
Даты
2024-07-11—Публикация
2023-12-14—Подача