Область техники, к которой относится изобретение
Настоящее изобретение относится к области биотехнологии для применения в сельском хозяйстве, в частности селекции и семеноводстве, а также генетике растений и может быть использовано для идентификации генотипов льна, обладающих различными хозяйственно-полезными признаками.
Уровень техники
Лен обыкновенный (Linum usitatissimum L.) является одним из древнейших растений, возделываемых человеком, семена которого традиционно используют для получения пищевого масла [Weiss Е., Zohary D., Hopf М. 2012. Domestication of Plants in the Old World - The Origin and Spread of Domesticated Plants in South-west Asia, Europe, and the Mediterranean Basin. Oxford University Press, New York]. Семена льна являются одним из богатейших растительных источников полезных для здоровья ω-3 жирных кислот, а также содержат ряд других полезных для человека соединений (прежде всего лигнаны), поэтому употребление продуктов из семян льна препятствует возникновению и развитию сердечно-сосудистых, онкологических и других заболеваний [Muir A.D., Westcott N.D. 2003. Flax: The genus Linum. CRC Press, London; Goyal A., Sharma V., Upadhyay N., et al. 2014. Flax and flaxseed oil: an ancient medicine & modern functional food. J Food Sci Technol. 51, 1633-1653; Imran M., Ahmad N., Anjum F.M., et al. 2015. Potential protective properties of flax lignan secoisolariciresinol diglucoside. Nutr J. 14, 71; Abdelhamid A.S., Martin N., Bridges C, et al. 2018. Polyunsaturated fatty acids for the primary and secondary prevention of cardiovascular disease. Cochrane Database Syst Rev. 7, CD012345; Kezimana P., Dmitriev A. A., Kudryavtseva A.V., et al. 2018. Secoisolariciresinol Diglucoside of Flaxseed and Its Metabolites: Biosynthesis and Potential for Nutraceuticals. Front Genet. 9, 641; Locke A., Schneiderhan J., Zick S.M. 2018. Diets for Health: Goals and Guidelines. Am Fam Physician. 97, 721-728; Parikh M., Netticadan Т., Pierce G.N. 2018. Flaxseed: its bioactive components and their cardiovascular benefits. Am J Physiol Heart Circ Physiol. 314, H146-H159; Campos J.R., Severino P., Ferreira C.S., et al. 2019. Linseed Essential Oil - Source of Lipids as Active Ingredients for Pharmaceuticals and Nutraceuticals. Curr Med Chem. 26, 4537-4558; Cullis C.A. 2019. Genetics and Genomics of Linum. Springer, Cham; Mali A.V., Padhye S.B., Anant S., et al. 2019. Anticancer and antimetastatic potential of enterolactone: Clinical, preclinical and mechanistic perspectives. Eur J Pharmacol. 852, 107-124]. Семена масличного льна применяются в производстве экологически безопасных красок, линолеума, композитных материалов, пищевых продуктов, биологически активных добавок, фармацевтических препаратов, а также функциональных кормов для животных [Kouba М., Mourot J. 2011. A review of nutritional effects on fat composition of animal products with special emphasis on n-3 polyunsaturated fatty acids. Biochimie. 93, 13-17; Singh K.K., Mridula D., Rehal J., et al. 2011. Flaxseed: apotential source of food, feed and fiber. Crit Rev Food Sci Nutr. 51, 210-222; Corino C, Rossi R., Cannata S., et al. 2014. Effect of dietary linseed on the nutritional value and quality of pork and pork products: systematic review and meta-analysis. Meat Sci. 98, 679-688; Goyal A., Sharma V., Upadhyay N., et al. 2014. Flax and flaxseed oil: an ancient medicine & modern functional food. J Food Sci Technol. 51, 1633-1653; Campos J.R., Severino P., Ferreira C.S., et al. 2019. Linseed Essential Oil - Source of Lipids as Active Ingredients for Pharmaceuticals and Nutraceuticals. Curr Med Chem. 26, 4537-4558; Fombuena V., Petrucci R., Dominici F., et al. 2019. Maleinized Linseed Oil as Epoxy Resin Hardener for Composites with High Bio Content Obtained from Linen Byproducts. Polymers (Basel). 11, 301].
Однонуклеотидный полиморфизм (SNP) представляет собой однонуклеотидную вариацию в одном нуклеотиде ДНК, которая встречается в определенном положении в геноме [Vignal, A., Milan, D., SanCristobal, М. & Eggen, A. A review on SNP and other types of molecular markers and their use in animal genetics. Genet. Selection Evolution, 34,3 34, 275-305 (2002).]. Широкое распространение SNP в гене или в регуляторной области обычно действует как биологический маркер для идентифицированных генов, ассоциированных с важными признаками [Yearn, I. Current advances and prospectus of viral resistance in horticultural crops. Horticulture, Environment, Biotechnol. 57, 113-122, (2016).]. У растений SNP имеют особенно важное применение, отражая как естественную генетическую изменчивость, так и генетический дрейф, созданный селекционерами во время улучшения растений [Shavrukov, Y. Comparison of SNP and CAPS markers application in genetic research in wheat and barley. Bmc Plant. Biol. 16, 11 (2016); Thomson, M. J. High-throughput SNP genotypingto accelerate crop improvement. Plant. Breed. Biotechnol. 2, 195-212 (2014)]. Таким образом, SNP являются идеальными маркерами для анализа генетического фона из-за их преимуществ в высокой пропускной способности обнаружения и легкой интеграции данных генотипирования [Tian, Н. L. et al. Development of maizeSNP3072, a high-throughput compatible SNP array, for DNA fingerprinting identification of Chinese maize varieties. Mol. Breed. 35, 136 (2015).].
В генетических исследованиях обычно используются несколько традиционных методов генотипирования SNP, основанных на системах электрофореза, таких как CAPS (расщепленная амплифицированная полиморфная последовательность), dCAPs (производный CAPS) и AS-PCR (аллель-специфическая ПЦР). В настоящее время очень широко используется технология секвенирования следующего поколения (NGS), с ее помощью собраны полногеномные последовательности сотен видов, что способствовало крупномасштабному повторному секвенированию и получению миллионов SNP для генетических исследований [Meng, K. et al. Development of SNP markers using RNA-seq technology and tetraprimer ARMS-PCR in sweetpotato. J. Integr. Agriculture 16, 464-470 (2017)].
Непрерывный прогресс в высокопроизводительном секвенировании облегчил разработку некоторых высокопроизводительных методов генотипирования SNP, таких как микрочипы [Semagn, K., Babu, R., Hearne, S. & Olsen, M. Single nucleotide polymorphism genotyping using Kompetitive Allele Specific PCR (KASP): overview of the technology and its application in crop improvement. Mol. Breed. 33, 1-14 (2014).]. Однако платформы на основе чипов требуют огромного количества времени для разработки SNP панелей и подходят только для генотипирования сотен образцов с тысячами SNP. Кроме того, огромные затраты на единовременные инвестиции в оборудование или микрочипы также ограничивают использование таких подходов в большинстве лабораторий.
Следовательно, использование экономичной и высокоточной технологии генотипирования среднемасштабных (30-500 маркеров) полногеномных SNP решит обозначенные проблемы позволит проводить анализ отдельных растений, используя для выделения ДНК лишь несколько листьев, давая возможность выполнять другие исследования этого генотипа и получить с него семена, а также различать искомые полиморфизмы в гетерозиготном и гомозиготном состояниях, что существенно повышает точность анализа и позволяет выполнять отбор ценных генотипов, начиная с ранних этапов селекционного процесса (например, с поколения F2 гибридов).
Использование ДНК-маркеров
Таким образом, разработка надежной и удобной в применении ДНК-тест-системы для идентификации полиморфизмов, отвечающих за хозяйственно-ценные признаки, откроет новые возможности для маркер-ориентированной селекции льна с целью создания сортов пищевого, фармацевтического или промышленного назначения, а также контроля сортовой чистоты при ведении семеноводства.
Раскрытие сущности изобретения
Целью создания изобретения являлась разработка ДНК-тест-системы, которая позволяет идентифицировать одновременно идентифицировать 32 локуса в геноме льна, ассоциированных с хозяйственно-ценными признаками. В таблице 1 приведены характеристики ДНК-маркеров и их ассоциация с хозяйственно-ценными признаками льна [Povkhova, L. V., Melnikova, N. V., Rozhmina, Т. A., Novakovskiy, R. О., Pushkova, Е. N., Dvorianinova, E. M., Zhuchenko, A. A., Kamionskaya, A. M., Krasnov, G. S. & Dmitriev, A. A. (2021) Genes Associated with the Flax Plant Type (Oil or Fiber) Identified Based on Genome and Transcriptome Sequencing Data, Plants. 10.; You, F. M., Xiao, J., Li, P., Yao, Z., Jia, G., He, L., Kumar, S., Soto-Cerda, В., Duguid, S. D., Booker, H. M., Rashid, K. Y. & Cloutier, S. (2018) Genome-Wide Association Study and Selection Signatures Detect Genomic Regions Associated with Seed Yield and Oil Quality in Flax, International journal of molecular sciences. 19.; You, F. M. & Cloutier, S. (2020) Mapping Quantitative Trait Loci onto Chromosome-Scale Pseudomolecules in Flax, Methods and protocols. 3.; Xie, D., Dai, Z., Yang, Z., Sun, J., Zhao, D., Yang, X., Zhang, L., Tang, Q. & Su, J. (2017) Genome-Wide Association Study Identifying Candidate Genes Influencing Important Agronomic Traits of Flax (Linum usitatissimum L.) Using SLAF-seq, Frontiers in plant science. 8, 2232; Xie, D., Dai, Z., Yang, Z., Tang, Q., Sun, J., Yang, X., Song, X., Lu, Y., Zhao, D., Zhang, L. & Su, J. (2018) Genomic variations and association study of agronomic traits in flax, BMC genomics. 19, 512; Kumar, S., You, F. M., Duguid, S., Booker, H., Rowland, G. & Cloutier, S. (2015) QTL for fatty acid composition and yield in linseed (Linum usitatissimum L.), TAG Theoretical and applied genetics Theoretische und angewandte Genetik. 128, 965-84; Dmitriev, A. A., Kezimana, P., Rozhmina, T. A., Zhuchenko, A. A., Povkhova, L. V., Pushkova, E. N., Novakovskiy, R. O., Pavelek, M., Vladimirov, G. N., Nikolaev, E. N, Kovaleva, O. A., Kostyukevich, Y. I., Chagovets, V. V., Romanova, E. V., Snezhkina, A. V., Kudryavtseva, A. V., Krasnov, G. S. & Melnikova, N. V. (2020) Genetic diversity of SAD and FAD genes responsible for the fatty acid composition in flax cultivars and lines, BMC plant biology. 20, 301; Kezimana, P., Rozhmina, T. A., Krasnov, G. S., Povkhova, L. V., Novakovskiy, R. O., Pushkova, E. N., Zhuchenko, A. A., Bjelkova, M., Pavelek, M., Dmitriev, A. A. & Melnikova, N. V. (2021). Evaluation of polymorphism of SAD and FAD genes in flax (Linum usitatissimum L.) cultivars and lines using deep sequencing. Paper presented at the Theory and practice of adaptive plant breeding (Zhuchenkov readings VI) Collection of scientific papers based on the materials of the International Scientific and Practical Conference, Krasnodar.].

Настоящее изобретение относится к новому способу идентификации генотипов льна обладающих хозяйственно-ценными признаками, который представляет собой мультиплексной амплификации 32 участков генома льна исследуемых образцов с последующим анализом продуктов амплификации с помощью технологии массивного параллельного секвенирования и сравнения полученных последовательностей с референсными аллелями.
Способ определения ДНК-маркеров включает следующие стадии:
а) получение материала растений льна (например, несколько листьев);
б) выделение ДНК;
в) проведение реакции мультиплексной амплификации 32 участков генома льна, несущих сайты СР027619.1:4809251, СР027619.1:5296528, СР027619.1:10459658, СР027619.1:16726604, СР027619.1:18443786, СР027619.1:25260635, СР027621.1:17365579, СР027622.1:1035355, СР027623.1:15861094, СР027624.1:1082928, СР027624.1:15460619, СР027625.1:1935390, СР027625.1:1939072, СР027625.1:11374706, СР027626.1:5926678, СР027626.1:17310210, СР027626.1:22402660, СР027626.1:22756656, СР027627.1:3929632, СР027627.1:3929632, СР027627.1:6245953, СР027627.1:7262787, СР027627.1:19668773, СР027627.1:3929632, СР027627.1:24655347, СР027629.1:16437137, СР027630.1:5995854, СР027630.1:14198976, СР027630.1:13776301, СР027631.1:12047740, СР027631.1:16092048, СР027633.1:3857714 в сборке генома льна GCA_000224295.2/ASM22429v2 с использованием смеси ген-специфичных праймеров и матрицы ДНК, полученной на стадии б;
г) проведение второго раунда реакции амплификации для введения в последовательность ампликона уникальных индексов, характеризующих данный образец, и универсальных последовательностей для последующего массивного параллельного секвенирования, с использованием смеси специфичных праймеров с уникальными для каждого образца индексами и матрицы ДНК, полученной на стадии в;
д) массивное параллельное секвенирование полученных на г) ампликонов
е) биоинформатический анализ полученных на стадии д) данных, в том числе отнесение каждого прочтения к конкретному образцу, сравнение полученных последовательностей с референсными аллелями и определение наличия полиморфизмов в них, а также их состояния - гомо- и гетерозиготного.
В еще одном воплощении заявленного способа в качестве стандартов на стадии е) используют последовательности ДНК SEQ ID NO: 65-96 (Перечень последовательностей).
Краткое описание фигур и таблиц Фигура 1. Схема проведения мультиплексного анализа определения наличия полиморфизмов в 32 локусах генома льна.
Осуществление изобретения
Данное изобретение предлагает способ идентификации 32 генотипов льна, ответственных за различные хозяйственно-полезные признаки, основанный мультиплексной амплификации 32 локусов генома льна, массивном параллельном секвенировании полученных ампликонов и сравнения полученных последовательностей с референсными последовательностями данных локусов.
Образцы тканей для анализа
В качестве материала для анализа могут быть использованы различные ткани растения льна, однако предпочтительным вариантом является использование нескольких листьев, что дает возможность выполнить другие исследования этого генотипа и получить с растения семена.
Выделение ДНК
Способы выделения ДНК из тканей растений хорошо известны и, как правило, включают следующие стадии: измельчение растительного материала, лизис клеток, выделение ДНК и ее очистку, проверку качества ДНК электрофорезом в 1%-ном агарозном геле в присутствии красителя бромистого этидия или его аналогов, а также спектрофотометрическое определение количества ДНК. Гомогенизацию материала можно проводить вручную с использованием пестиков или с помощью гомогенизаторов, например, MagNA Lyser (Roche, Швейцария) или TissueLyser II (Qiagen, Германия) в пробирках с керамическими, стеклянными или стальными шариками. Для выделения ДНК могут быть использованы различные протоколы или наборы реактивов. В классических методах выделения ДНК из растений в качестве лизирующего агента используется буфер, содержащий гуанидин хлорид или ЦТАБ (цетилтриметиламмоний бромид) с последующей экстракцией хлороформом. Для выделения ДНК из растений также доступны коммерческие наборы, например, ДНК-Экстран-3 (Синтол, Россия) и DNeasy Plant Mini Kit (Qiagen, Германия).
Выбор специфичных праймеров
Выбор специфичных праймеров осуществлялся с использованием полученных авторами и другими исследователями данных о полиморфизмах 32 локусов, а также программ NCBI Primer-BLAST [Ye J., Coulouris G., Zaretskaya I., et al. 2012. Primer-BLAST: a tool to design target-specific primers for polymerase chain reaction. BMC Bioinformatics. 13, 134] и MethyMer [Krasnov G.S., Melnikova N.V., Lakunina V.A., et al. 2018. MethyMer: Design of combinations of specific primers for bisulfite sequencing of complete CpG islands. J Bioinform Comput Biol. 16, 1840004]. Подбор праймеров для амплификации локусов, в которых локализован полиморфизм, осуществляли с целью дальнейшего анализа методом массивного параллельного секвенирования ампликонов, проводился в соответствии со стандартными рекомендациями и требованиями метода. В предпочтительном воплощении используются праймеры SEQ ID NO: 1-64 (Перечень последовательностей).
Определение полиморфизмов в 32 локусах генома льна
Метод массивного параллельного секвенирования широко применяется для идентификации ДНК-полиморфизмов и генотипирования образцов [Davey, J., Hohenlohe, P., Etter, P. et al. Genome-wide genetic marker discovery and genotyping using next-generation sequencing. Nat Rev Genet 12, 499-510 (2011); Nielsen R, Paul JS, Albrechtsen A, Song YS. Genotype and SNP calling from next-generation sequencing data. Nat Rev Genet. 2011 Jun;12(6):443-51]. В настоящем изобретении для образцов растений льна используется сравнительный анализ полученных последовательностей локусов, полученных в результате их мультиплексной амплификации с помощью праймеров SEQ ID NO: 1-64 (Перечень последовательностей) и массивного параллельного секвенирования ампликонов, с референсными последовательностями локусов SEQ ID NO: 65-96 (Перечень последовательностей).
Далее настоящее изобретение детально проиллюстрировано с использованием конкретных примеров, представляющих предпочтительные воплощения изобретения. В то же время, изобретение не ограничивается описанными воплощениями, а включает любые альтернативные варианты и модификации, применение которых возможно с учетом сущности изобретения.
Пример 1. Подготовка образцов
Семена сортов и линий льна обрабатывают 3% раствором гипохлорита натрия, промывают дистиллированной водой и проращивают в чашках Петри на фильтровальной бумаге, смоченной водой. Для выделения ДНК используют листья 7-дневных проростков.
Пример 2. Выделение ДНК
Листья измельчают на гомогенизаторе TissueLyser II (Qiagen, Германия), после чего проводят экстракцию ДНК набором ДНК-Экстран-3 (Синтол, Россия) согласно протоколу производителя. Качество ДНК оценивают методом электрофореза в 1%-ном агарозном геле. Концентрацию ДНК определяют на флуориметре Qubit (Thermo Fisher Scientific, США) или спектрофотометре NanoDrop (Thermo Fisher Scientific).
Пример 3. Мультиплексная амплификация 32 локусов генома льна и анализ с помощью массивного параллельного секвенирования.
1. Готовят реакционную смесь для амплификации, смешивая все компоненты реакции, за исключением ДНК, из расчета 1 реакция объемом 20 мкл (из которых 2 мкл отведено под ДНК) для каждого образца и стандарта плюс 10%. Состав реакционной смеси для мультиплексной амплификации следующий: 1x Taq Turbo буфер (Евроген, Россия), 250 нМ каждого дезоксинуклеозидтрифосфата (Евроген), 100 нМ каждого из 64 праймеров (SEQ ID NO: 1-64), 2 ед. HS Taq ДНК-полимер азы (Евроген).
2. В лунки ПЦР-плашки вносят по 18 мкл приготовленной реакционной смеси без ДНК.
3. Вносят в лунки ПЦР-плашки по 2 мкл ДНК (концентрация 12,5 нг/мкл для образцов), плотно заклеивают плашку пленкой.
4. Помещают плашку в приборное отделение амплификатора, задают названия ячеек и программу: +95°С - 10 мин - первичная денатурация ДНК; 40 циклов амплификации: +95°С - 15 сек - денатурация, +60°С - 20 сек - отжиг праймеров, +72°С мин элонгация цепи ДНК.
5. Проводят очистку продуктов амплификации с помощью набора Cleanup Standard (Евроген, Россия) согласно протоколу производителя.
6. Проводят второй раунд амплификации для введения в последовательности ампликонов уникальных индексов образца и универсальных последовательностей для секвенирования проводили с помощью ПЦР, смешивая все компоненты реакции, за исключением ДНК, из расчета 1 реакция объемом 20 мкл (из которых 5 мкл отведено под ДНК) для каждого образца и стандарта плюс 10%. Состав реакционной смеси для мультиплексной амплификации следующий: 1x Taq Turbo буфер (Евроген, Россия), 250 нМ каждого дезоксинуклеозидтрифосфата (Евроген), 250 нМ каждого из праймеров (SEQ ID NO: 97 и 98), 2 ед. HS Taq ДНК-полимеразы (Евроген).
7. В лунки ПЦР-плашки вносят по 15 мкл приготовленной реакционной смеси без ДНК.
8. Вносят в лунки ПЦР-плашки по 5 мкл ДНК из шага 5), плотно заклеивают плашку пленкой.
9. Помещают плашку в приборное отделение амплификатора, задают названия ячеек и программу: +95°С 10 мин первичная денатурация ДНК; 30 циклов амплификации: +95°С - 15 сек - денатурация, +60°С - 20 сек - отжиг праймеров, +72°С мин - элонгация цепи ДНК.
10. Проводят очистку продуктов амплификации с помощью набора Cleanup Standard (Евроген, Россия) согласно протоколу производителя.
11. Проводят секвенирование ампликонов на приборе MiSeq Illumina с помощью набора v3, 1 x 150 п. о. согласно протоколу производителя.
12. Проводят обработку данных путем сравнительного выравнивания полученных последовательностей с референсной последовательностью SEQ ID NO: 65-96 (Перечень последовательностей). Проводят статистический анализ выявленных изменений в нуклеотидной последовательности локуса для определения гомо- и гетерозиготного состояния аллеля.
Приведенное выше подробное описание изобретения и его конкретных воплощений, представленных в примерах, предназначено исключительно для более полного уяснения сущности заявленного изобретения, но не для его ограничения. Могут быть внесены изменения, которые, однако, будут соответствовать сущности настоящего изобретения, определяющейся прилагаемой формулой изобретения.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Способ
мультиплексной идентификации 32 генетических маркеров льна.xml"
softwareName="WIPO Sequence" softwareVersion="2.2.0"
productionDate="2023-04-17">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2022135242/20(076436)</ApplicationNumberText>
<FilingDate></FilingDate>
</ApplicationIdentification>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2022135242/20(076436)</ApplicationNumberText>
<FilingDate></FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное учреждение науки Институт общей генетики им. Н.И. Вавилова
Российской академии наук (ИОГен РАН)</ApplicantName>
<ApplicantNameLatin>Vavilov Institute of General Genetics
RAS</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ мультиплексной
идентификации 32 генетических маркеров льна</InventionTitle>
<SequenceTotalQuantity>98</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgctcgacttgagtaggatctc</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>48</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..48</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q25">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccaggatgatcaagggtatgatttg<
/INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>46</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgctagtgcagaactggtgtatg</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>49</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..49</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q27">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccactacattaacagaggagtgaagg
</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgcctgaccaaaatcctggctc</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>51</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..51</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q29">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccagtactgaaaatatgtattggatt
gg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccggttttcttatcgcttctgtc</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>49</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..49</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q32">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccagacgttgtccagcgaataatatg
</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>46</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccggtacatgtgaacagcgtattg</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q36">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccacaactcaacttcaacggtatg</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>48</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..48</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q38">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgcaatttccatcctactctctgtg<
/INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q40">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccacacactgccattgtagtaagc</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q42">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccggcacacgacatcacatgtattg</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>48</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..48</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q44">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccagtttcatataccatcttccatc<
/INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>46</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q54">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgtttatctgggttgttgcatgc</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q56">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccaggtacgaggattgaggaatgg</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q58">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgttgaaaactgtggcctttcc</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>46</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q60">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccacggttggggagatgattaag</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q62">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgagtacgagaagtgtagacag</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>46</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q64">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccagaacccaacttggcagattg</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q66">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgccctttctctgcatggtttctg</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q68">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccacctcagtcttttgctgaacag</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="23">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q70">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgaggcagatggcagggatcaa</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="24">
<INSDSeq>
<INSDSeq_length>46</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q72">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccactccaaaggcttgcttacac</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="25">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q74">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgtaaacccatccgtgaacttg</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="26">
<INSDSeq>
<INSDSeq_length>48</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..48</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q76">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccagtcgagatccattttcgaatcg<
/INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="27">
<INSDSeq>
<INSDSeq_length>44</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q78">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccggctttggttgaggtggatg</INS
DSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="28">
<INSDSeq>
<INSDSeq_length>48</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..48</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q80">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccattacggttattcgtgatctacc<
/INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="29">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q82">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgttccaggtcagttcctgagg</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="30">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q84">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccatgtgagtgcaaagaaaacctg</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="31">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q86">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgatcatggcgccacttgtgtc</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="32">
<INSDSeq>
<INSDSeq_length>50</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q88">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccatctggaactctaaacccttagaa
g</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="33">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q90">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgactatgggtatacctggctg</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="34">
<INSDSeq>
<INSDSeq_length>46</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q92">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccactcaacgcaaatcccaaacg</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="35">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q94">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgtgcttgctggtaaagccatc</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="36">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q96">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccaccatcccacttcattcctcag</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="37">
<INSDSeq>
<INSDSeq_length>46</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q98">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgtgtggcatgtaaatggccttg</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="38">
<INSDSeq>
<INSDSeq_length>46</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q100">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccaagagccgagttgaatgacga</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="39">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q102">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgtaggctttggctgaaaggtg</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="40">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q104">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccaaggtggtttttagcaatgacg</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="41">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q106">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgcagtgttggcttttcacctg</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="42">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q108">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccactgcaacataacaaggagagc</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="43">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q110">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgcctcgccatacctattgttg</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="44">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q112">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccatggaaccctaaacctgttgac</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="45">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q114">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgaatgtcatccccacagactc</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="46">
<INSDSeq>
<INSDSeq_length>51</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..51</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q116">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccaggtttgtgtatttttgcctatca
ac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="47">
<INSDSeq>
<INSDSeq_length>46</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q118">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccggatgccagagggaagaactac</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="48">
<INSDSeq>
<INSDSeq_length>46</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q120">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccagacatgcaacccaccctttg</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="49">
<INSDSeq>
<INSDSeq_length>46</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q122">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgcgctcgatagtcggttagatc</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="50">
<INSDSeq>
<INSDSeq_length>46</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q124">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccaggctgcccaatgaatgtatg</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="51">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q126">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgactgggattgtttgcccttg</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="52">
<INSDSeq>
<INSDSeq_length>46</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q128">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccacattcacatggcattggtcc</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="53">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q130">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgtacccgatgacaagtcgatc</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="54">
<INSDSeq>
<INSDSeq_length>46</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q132">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccagtggtgcttcatatgtggtc</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="55">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q134">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccggtcattttcggtgcttccac</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="56">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q136">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccataacctcgtttatgtgccaag</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="57">
<INSDSeq>
<INSDSeq_length>46</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q138">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccggccatggtttggttactctgg</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="58">
<INSDSeq>
<INSDSeq_length>46</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q140">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccatcgccatatatagcagcacc</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="59">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q142">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgctagagaagggttagggttgga</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="60">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q144">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccagaaaatctccaccctccgatc</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="61">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q146">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgcaaagactgcccagaacatg</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="62">
<INSDSeq>
<INSDSeq_length>46</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q148">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccatcactcagctacgtcctgag</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="63">
<INSDSeq>
<INSDSeq_length>48</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..48</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q150">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgaccagtacacagaaaaggattcc<
/INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="64">
<INSDSeq>
<INSDSeq_length>49</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..49</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q152">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttccttggcacccgagaattccactcttcccatctgctagaagaac
</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="65">
<INSDSeq>
<INSDSeq_length>146</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..146</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q221">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgctcgacttgagtaggatctccgag
aggggaaacataagacatccaataacgcccaatgcactctccattaacccaaatcatacccttgatcatc
ctggaattctcgggtgccaaggaactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="66">
<INSDSeq>
<INSDSeq_length>178</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..178</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q222">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgctagtgcagaactggtgtatgttt
ctgtaatgtttgttaagttatgtccctagtggtcgttaatgttactgtacttatctgttcttctccatcg
agccaacataccttcactcctctgttaatgtagtggaattctcgggtgccaaggaactc</INSDSeq_s
equence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="67">
<INSDSeq>
<INSDSeq_length>204</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..204</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q223">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgcctgaccaaaatcctggctccgcc
cctacatttatattatattttactaaaagcagtacaatgaatttatttttagatttaatttatcaattaa
ttactcaatacatacataaatttttaatttaaatccaatccaatacatattttcagtactggaattctcg
ggtgccaaggaactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="68">
<INSDSeq>
<INSDSeq_length>157</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..157</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q224">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccggttttcttatcgcttctgtcgttg
gatatcaagaactgaataaaaaaaataaaaaagtttactccatatatttcaaaactcctcatattattcg
ctggacaacgtctggaattctcgggtgccaaggaactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="69">
<INSDSeq>
<INSDSeq_length>124</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..124</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q225">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccggtacatgtgaacagcgtattgatg
ttttttctacatactcattatgtttattcataccgttgaagttgagttgtggaattctcgggtgccaagg
aactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="70">
<INSDSeq>
<INSDSeq_length>165</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..165</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q226">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgcaatttccatcctactctctgtgt
ttctgtctctgttttcactgcatactctgttttctctgcaacacttagaaaaaaaacagagagtatgggg
cttactacaatggcagtgtgtggaattctcgggtgccaaggaactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="71">
<INSDSeq>
<INSDSeq_length>199</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..199</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q227">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccggcacacgacatcacatgtattgtt
taacagttcatctaaccaatctactaagtactatatcttcaataatatgattatccacggcgaattttct
ccgaagtagaagttggtatgtcatgtaaaatagatggaagatggtatatgaaactggaattctcgggtgc
caaggaactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="72">
<INSDSeq>
<INSDSeq_length>152</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..152</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q228">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgtttatctgggttgttgcatgcagt
ggccatgggagcttctcagacatctggttgttgaacaatgtgatgggacatatactccattcctcaatcc
tcgtacctggaattctcgggtgccaaggaactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="73">
<INSDSeq>
<INSDSeq_length>141</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..141</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q229">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgttgaaaactgtggcctttccttta
cctcctcctccaacaacaactctccttctccactcttcgacggcggcttaatcatctccccaaccgtgga
attctcgggtgccaaggaactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="74">
<INSDSeq>
<INSDSeq_length>197</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..197</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q230">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgagtacgagaagtgtagacagaact
tctctgacaaacccacaaaacgtttgcatggagaacgacaatagtaaagagttttaacatgtaaaaacac
caaatatacatagccccatccatacattttaccaatctgccaagttgggttctggaattctcgggtgcca
aggaactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="75">
<INSDSeq>
<INSDSeq_length>170</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..170</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q231">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgccctttctctgcatggtttctgta
tgttcttattggaatagagtccaccatgaacctgatcacagattaaacacatattttcattaattgtgag
aaaactgttcagcaaaagactgaggtggaattctcgggtgccaaggaactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="76">
<INSDSeq>
<INSDSeq_length>140</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..140</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q232">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgaggcagatggcagggatcaagcag
taggcgataagaggcaaggaagtgaaagggtaaacaatggtgttggtgtaagcaagcctttggagtggaa
ttctcgggtgccaaggaactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="77">
<INSDSeq>
<INSDSeq_length>192</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..192</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q233">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccggtcgagatccattttcgaatcgga
tgtcaaatctaagaaaggggtttataccgcttttgatattttgggaactcaaaatgacgagttctgcaat
tagtacagtttgtatcggacaaaaaatcaagttcacggatgggtttatggaattctcgggtgccaaggaa
ctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="78">
<INSDSeq>
<INSDSeq_length>197</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..197</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q234">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccggctttggttgaggtggatgaacga
ggcatggcggcggtgatgagttttgcggcgatggtgttataaaaaatttaaaggtgaaaactgaaaagcc
ccaaaataggagagaaaaaccctaacttaaggtagatcacgaataaccgtaatggaattctcgggtgcca
aggaactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="79">
<INSDSeq>
<INSDSeq_length>215</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..215</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q235">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgtgtgagtgcaaagaaaacctgaaa
cattaatttataagccatatatgttagtacttctgaatcaaaagaactaaataacaggttatatttacaa
ataactagttataattaatcctctataactagttaataattatttataatcctcaggaactgacctggaa
tggaattctcgggtgccaaggaactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="80">
<INSDSeq>
<INSDSeq_length>104</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..104</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q236">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgatcatggcgccacttgtgtctacc
gtagtcttctaagggtttagagttccagatggaattctcgggtgccaaggaactc</INSDSeq_seque
nce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="81">
<INSDSeq>
<INSDSeq_length>158</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..158</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q237">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgactatgggtatacctggctggaag
tttaggtatcgggtaatcccgatcccgcgaattcgagttgggtttgggtatccgatatccgtacgtttgg
gatttgcgttgagtggaattctcgggtgccaaggaactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="82">
<INSDSeq>
<INSDSeq_length>185</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..185</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q238">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgtgcttgctggtaaagccatcttct
tttttttccgctctcccctatattcacatagatcctcaacattcttatggagattctaatgtttcattat
gatgtttaattcagcaattctgaggaatgaagtgggatggtggaattctcgggtgccaaggaactc</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="83">
<INSDSeq>
<INSDSeq_length>226</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..226</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q239">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgagagccgagttgaatgacgatacg
aatccaaaaatacacccatctatatatattacgttagtattatttaataataaaagataaaaaaaaatga
acatgcaacgtgctgtgttttgttgcaaagaaattgaagaagagaataagaagagaattccaaggccatt
tacatgccacatggaattctcgggtgccaaggaactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="84">
<INSDSeq>
<INSDSeq_length>175</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..175</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q240">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgaggtggtttttagcaatgacgatg
tgaatatgatatatccatcttcgttaacaaatggaaacttaaatatgaaaaaattaaatgaatgagactg
acccacgtatcacctttcagccaaagcctatggaattctcgggtgccaaggaactc</INSDSeq_sequ
ence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="85">
<INSDSeq>
<INSDSeq_length>177</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..177</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q241">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgctgcaacataacaaggagagcatt
ataatttataaacacattgaaaacatgaacgataatctaattggaaggcaagagtaaaaagggtttagtt
acctccacatctcaggtgaaaagccaacactgtggaattctcgggtgccaaggaactc</INSDSeq_se
quence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="86">
<INSDSeq>
<INSDSeq_length>176</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..176</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q242">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgcctcgccatacctattgttgatgc
atgtttaaggttttgcaattagatttcaattgaagttataaaatacatcaacgtagcatgattgtatgtg
acaagaagaagtcaacaggtttagggttccatggaattctcgggtgccaaggaactc</INSDSeq_seq
uence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="87">
<INSDSeq>
<INSDSeq_length>181</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..181</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q243">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgaatgtcatccccacagactctgca
cgccttgggaggagcagaagactggcgggtggcaggccgctgctgctgctgttaagaatcacagaggcat
ttcatcaaacagttgataggcaaaaatacacaaacctggaattctcgggtgccaaggaactc</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="88">
<INSDSeq>
<INSDSeq_length>153</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..153</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q244">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccggacatgcaacccaccctttgtata
gtatagtggcctcaaatcgttctcgtggatatgtaacatagatgctgctaattccttgtagttcttccct
ctggcatctggaattctcgggtgccaaggaactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="89">
<INSDSeq>
<INSDSeq_length>163</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..163</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q245">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgagtagccgttggagactacctata
acgcgccacgcgtcctaggatgccccgcgtgttgtaggcagcctccaacgaattctataaatatccctcc
tccctttcttcattcctctggaattctcgggtgccaaggaactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="90">
<INSDSeq>
<INSDSeq_length>197</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..197</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q246">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgactgggattgtttgcccttgatta
aagttttgatcttttcaatccatatgttgggaaaatcgattaaacagttgtctttttggcgaaatttaca
atgtctttgtttggctactgagaagtacgaggggaccaatgccatgtgaatgtggaattctcgggtgcca
aggaactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="91">
<INSDSeq>
<INSDSeq_length>141</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..141</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q247">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgtacccgatgacaagtcgatcgaag
atgttcacaccaatgccttaacaccatccactgaccccaagacaaagaccacatatgaagcaccactgga
attctcgggtgccaaggaactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="92">
<INSDSeq>
<INSDSeq_length>136</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..136</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q248">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccggtcattttcggtgcttccacttca
cattttcaatgtaagtcaccatgcatgtttagacatataacttggcacataaacgaggttatggaattct
cgggtgccaaggaactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="93">
<INSDSeq>
<INSDSeq_length>142</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..142</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q249">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccggccatggtttggttactctggtta
tttgattcaaacagagatgggtttggtgagtgatgaggcagggggtaggtgctgctatatatggcgatgg
aattctcgggtgccaaggaactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="94">
<INSDSeq>
<INSDSeq_length>133</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..133</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q250">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgctagagaagggttagggttggatg
gaatgggttcaaatagaagtaagagttgttggcaacagatcggagggtggagattttctggaattctcgg
gtgccaaggaactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="95">
<INSDSeq>
<INSDSeq_length>181</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..181</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q214">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgcaaagactgcccagaacatggttc
cttgagcaatccagtagagaggccagacagtccagctgtccagctttgtcgccgcaacggcgaagctgag
gatgaccaggaggtctctcaggacgtagctgagtgatggaattctcgggtgccaaggaactc</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="96">
<INSDSeq>
<INSDSeq_length>254</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..254</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q216">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Linum usitatissimum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctttccctacacgacgctcttccgaccagtacacagaaaaggattccc
aacaaaaattagagaagatacactgatagtttacacattaatgggtaaagaaaggaaataacaaccaaaa
agcagttcacttttacatcagataataatagtttgtttgcaggaaaatacaaaaagattacatcggggta
attcatgcttatatttgttcttctagcagatgggaagagtggaattctcgggtgccaaggaactc</INS
DSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="97">
<INSDSeq>
<INSDSeq_length>54</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..54</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q218">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aatgatacggcgaccaccgagatctacactctttccctacacgacgctc
ttccg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="98">
<INSDSeq>
<INSDSeq_length>59</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..59</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q220">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caagcagaagacggcatacgagatnnnnnnnngtgactggagttccttg
gcacccgaga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ мультиплексной идентификации 52 генетических маркеров хозяйственно ценных признаков льна | 2023 |
|
RU2826718C1 |
| СПОСОБ ГЕНОТИПИРОВАНИЯ ИЗОЛЯТОВ NEISSERIA GONORRHOEAE НА БИОЛОГИЧЕСКОМ МИКРОЧИПЕ | 2023 |
|
RU2816767C1 |
| Нуклеотидная последовательность, кодирующая фермент литиказу, и панель олигонуклеотидов для получения синтетической нуклеотидной последовательности гена литиказы | 2023 |
|
RU2826150C1 |
| СПОСОБ ОПРЕДЕЛЕНИЯ СУБЛИНИЙ ВОЗБУДИТЕЛЯ ТУБЕРКУЛЕЗА ЛИНИИ L2 Beijing НА БИОЛОГИЧЕСКИХ МИКРОЧИПАХ | 2022 |
|
RU2790296C1 |
| СПОСОБ ИДЕНТИФИКАЦИИ ОДНОНУКЛЕОТИДНЫХ ПОЛИМОРФИЗМОВ В МНОГОКОПИЙНОМ ЛОКУСЕ ГЕНОМА МЕТОДОМ ПЕТЛЕВОЙ ПОЛИМЕРАЗНОЙ ЦЕПНОЙ РЕАКЦИИ С ПОСЛЕДУЮЩЕЙ ГИБРИДИЗАЦИЕЙ НА БИОЛОГИЧЕСКОМ МИКРОЧИПЕ | 2022 |
|
RU2809184C1 |
| СПОСОБ ВЫДЕЛЕНИЯ ТОТАЛЬНОЙ ДНК БАКТЕРИЙ ИЗ ОБРАЗЦОВ ПОЧВЫ, СПОСОБ ОЦЕНКИ БАКТЕРИАЛЬНОГО СОСТАВА ПОЧВ ПОСРЕДСТВОМ МЕТАГЕНОМНОГО СЕКВЕНИРОВАНИЯ И НАБОРЫ ДЛЯ ОСУЩЕСТВЛЕНИЯ СПОСОБОВ | 2024 |
|
RU2829656C1 |
| СПОСОБ ВЫЯВЛЕНИЯ STREPTOCOCCUS MUTANS МЕТОДОМ ИЗОТЕРМИЧЕСКОЙ ПЕТЛЕВОЙ АМПЛИФИКАЦИИ | 2022 |
|
RU2799413C1 |
| НАБОР ПРАЙМЕРОВ ДЛЯ ВЫЯВЛЕНИЯ STREPTOCOCCUS MUTANS МЕТОДОМ ИЗОТЕРМИЧЕСКОЙ ПЕТЛЕВОЙ АМПЛИФИКАЦИИ | 2022 |
|
RU2799414C1 |
| НАБОР ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ ДЛЯ ГЕНОТИПИРОВАНИЯ ГРИБОВ ВИДОВОГО КОМПЛЕКСА COLLETOTRICHUM ACUTATUM | 2022 |
|
RU2807352C1 |
| Панель праймеров для полногеномного секвенирования вирусов парагриппа человека 1 и 2 типа | 2024 |
|
RU2833687C1 |
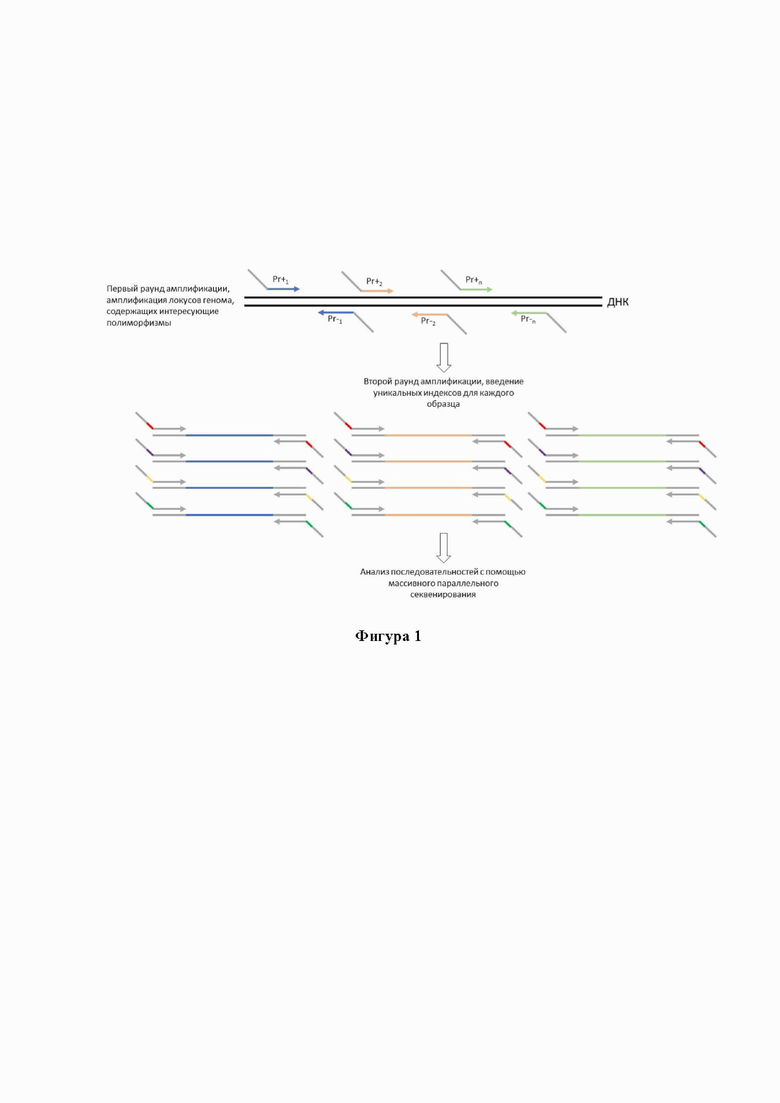
Изобретение относится к области молекулярной биологии. Описан способ параллельной идентификации 32 генетических маркеров льна, ассоциированных с различными хозяйственно полезными признаками, путем проведения мультиплексной амплификации 32 участков генома льна, несущих сайты CP027619.1:4809251, CP027619.1:5296528, CP027619.1:10459658, CP027619.1:16726604, CP027619.1:18443786, CP027619.1:25260635, CP027621.1:17365579, CP027622.1:1035355, CP027623.1:15861094, CP027624.1:1082928, CP027624.1:15460619, CP027625.1:1935390, CP027625.1:1939072, CP027625.1:11374706, CP027626.1:5926678, CP027626.1:17310210, CP027626.1:22402660, CP027626.1:22756656, CP027627.1:3929632, CP027627.1:3929632, CP027627.1:6245953, CP027627.1:7262787, CP027627.1:19668773, CP027627.1:3929632, CP027627.1:24655347, CP027629.1:16437137, CP027630.1:5995854, CP027630.1:14198976, CP027630.1:13776301, CP027631.1:12047740, CP027631.1:16092048, CP027633.1:3857714 в сборке генома льна GCA_000224295.2/ASM22429v2. Для это используют праймеры, представленные последовательностями SEQ ID NO: 1 - SEQ ID NO: 64, с последующим анализом продуктов амплификации с помощью технологии массивного параллельного секвенирования и сравнения полученных последовательностей с референсными аллелями, представленными последовательностями SEQ ID NO: 65-96. Заявленное изобретение позволяет одновременно идентифицировать 32 локуса в геноме льна, ассоциированных с хозяйственно-ценными признаками. 1 ил., 1 табл., 3 пр.
Способ параллельной идентификации 32 генетических маркеров льна, ассоциированных с различными хозяйственно полезными признаками, путем проведения мультиплексной амплификации 32 участков генома льна, несущих сайты CP027619.1:4809251, CP027619.1:5296528, CP027619.1:10459658, CP027619.1:16726604, CP027619.1:18443786, CP027619.1:25260635, CP027621.1:17365579, CP027622.1:1035355, CP027623.1:15861094, CP027624.1:1082928, CP027624.1:15460619, CP027625.1:1935390, CP027625.1:1939072, CP027625.1:11374706, CP027626.1:5926678, CP027626.1:17310210, CP027626.1:22402660, CP027626.1:22756656, CP027627.1:3929632, CP027627.1:3929632, CP027627.1:6245953, CP027627.1:7262787, CP027627.1:19668773, CP027627.1:3929632, CP027627.1:24655347, CP027629.1:16437137, CP027630.1:5995854, CP027630.1:14198976, CP027630.1:13776301, CP027631.1:12047740, CP027631.1:16092048, CP027633.1:3857714 в сборке генома льна GCA_000224295.2/ASM22429v2, с использованием праймеров, представленных последовательностями SEQ ID NO: 1 - SEQ ID NO: 64, с последующим анализом продуктов амплификации с помощью технологии массивного параллельного секвенирования и сравнения полученных последовательностей с референсными аллелями, представленными последовательностями SEQ ID NO: 65-96.
| ДИФФЕРЕНЦИРУЮЩИЙ И СПЕЦИФИЧЕСКИЙ ОЛИГОНУКЛЕОТИДЫ ДЛЯ ИДЕНТИФИКАЦИИ ПОСЛЕДОВАТЕЛЬНОСТЕЙ ДНК ТРАНСГЕННЫХ РАСТЕНИЙ В ПИЩЕВЫХ ПРОДУКТАХ, СПОСОБ ИДЕНТИФИКАЦИИ ТРАНСГЕННЫХ ПРОДУКТОВ, БИОЧИП, КОМБИНАЦИЯ ОЛИГОНУКЛЕОТИДОВ (ВАРИАНТЫ) И НАБОР ДЛЯ ОСУЩЕСТВЛЕНИЯ ЭТОГО СПОСОБА | 2007 |
|
RU2453605C2 |
| WO 2020099614 A1, 22.05.2020 | |||
| ИДЕНТИФИКАЦИЯ, ПРОИЗВОДСТВО И ПРИМЕНЕНИЕ НЕОАНТИГЕНОВ | 2016 |
|
RU2729116C2 |
| СПОСОБ ВЫЯВЛЕНИЯ РЕЦЕССИВНЫХ ФАКТОРОВ НАСЛЕДСТВЕННОЙ ПРЕДРАСПОЛОЖЕННОСТИ К РАКУ МОЛОЧНОЙ ЖЕЛЕЗЫ | 2013 |
|
RU2535157C1 |
Авторы
Даты
2023-10-09—Публикация
2022-12-29—Подача