Изобретение относится к области биотехнологии. Изобретение обеспечивает повышенную чувствительность клеток глиобластомы к темозоломиду с помощью подавления экспрессии энхансерной РНК. Изобретение также включает последовательность синтетической РНК для РНК-интерференции. Комбинация темозоломида и малой интерферирующей РНК, нацеленной на энхансерную РНК ENSG00000289579, может быть использована в противоопухолевой терапии.
Уровень техники
Глиобластома является наиболее частой и агрессивной опухолью головного мозга. На данный момент стандартный протокол лечения глиобластомы включает в себя прием алкилирующего агента темозоломида в сочетании с лучевой терапией и хирургическим удалением опухоли (Kotecha, R., Odia, Y., Khosla, A.A., & Ahluwalia, M.S. (2023). Key clinical principles in the management of glioblastoma. JCO Oncology Practice, 19(4), 180-189.). Но даже под воздействием такой комплексной терапии прогнозы по выживаемости остаются неутешительными для пациентов: медиана выживаемости составляет менее двух лет, а пятилетняя выживаемость менее 10 процентов (Tan et al. (2020). Management of glioblastoma: State of the art and future directions. CA: a cancer journal for clinicians, 70(4), 299-312). Такой плохой прогноз обусловлен, в частности, резистентностью к химиотерапии, приводящей к рецидивам опухоли после хирургического удаления. Было показано, что около половины опухолей устойчивы к темозоломиду (Lee (2016). Temozolomide resistance in glioblastoma multiforme. Genes & diseases, 3(3), 198-210).
В настоящее время идет активное исследование новых способов борьбы с устойчивостью к темозоломиду. Есть работы, которые показывают, что использование подходов, направленных на уменьшение экспрессии STAT3 методом РНК-интерференции (Kohsaka et al (2012). STAT3 inhibition overcomes temozolomide resistance in glioblastoma by downregulating MGMT expression. Molecular cancer therapeutics, 11(6), 1289-1299), или воздействие непосредственно на белок STAT3 ингибиторами (Chuang et al (2019). Preclinical evidence of STAT3 inhibitor pacritinib overcoming temozolomide resistance via downregulating miR-21-enriched exosomes from M2 glioblastoma-associated macrophages. Journal of Clinical Medicine, 8(7), 959), или ингибирование сигнального пути активации STAT3 (Yeom et al. (2014). RRAD promotes EGFR-mediated STAT3 activation and induces temozolomide resistance of malignant glioblastoma. Molecular cancer therapeutics, 13(12), 3049-3061), увеличивает чувствительность глиобластомы к темозоломиду. Еще одной мишенью, используемой для подавления развития глиобластомы, является белок mTOR. Было показано, что нокдаун и фармакологическое ингибирование рапамицином снижало экспрессию SOX2 и SOX9 и устраняло химиорезистентность (Garros-Regulez et al. (2016). mTOR inhibition decreases SOX2-SOX9 mediated glioma stem cell activity and temozolomide resistance. Expert opinion on therapeutic targets, 20(4), 393-405). Также использовали комбинацию ингибиторов STAT3 и mTOR для усиления эффекта от терапии и минимизации шанса возникновения устойчивой популяции (Miyata et al. (2017). Combination of a STAT3 inhibitor and an mTOR inhibitor against a temozolomide-resistant glioblastoma cell line. Cancer genomics & proteomics, 14(1), 83-91). Стоить отметить, что снижение активности вышеописанные белков воздействует не только на опухолевые, но и на здоровые клетки организма, что особенно сильно отражается на активно делящихся клетках (Chiba (2016). STAT3 inhibitors for cancer therapy-the rationale and remained problems. EC cancer, 1(S1), S1-S8). Поэтому особое значение имеют подходы, воздействующие не на распространенные в большинстве клеток белки, контролирующие клеточный рост и апоптоз, а на гены, регулирующие такие белки. Таким образом, побочные эффекты терапии проявляются только в клетках, которые зависят от экспрессии данных генов-регуляторов.
В последнее время все больший интерес вызывает клиническое применение терапевтических препаратов на основе РНК. Управлением по контролю качества пищевых продуктов и лекарственных средств США (FDA) и Европейским агентством по лекарственным средствам (ЕМА) одобрено 11 РНК-терапевтических препаратов, направленных на модификацию генов в печени, мышцах или центральной нервной системе. Все эти препараты представляют собой либо малые интерферирующие РНК (siRNA), либо антисмысловые олигонуклеотиды, вызывающие специфическую регуляцию генов, либо направленные на сплайсинг пре-мРНК (Winkle et al (2021). Noncoding RNA therapeutics - Challenges and potential solutions. Nature reviews Drug discovery, 20(8), 629-651). Вместе с этим появляется все больше данных о роли длинных некодирующих РНК (днРНК) в развитии различных заболеваний, особенно онкологических. Было показано, что днРНК также связаны с лекарственной устойчивостью в раке (Jiang et al. (2020). Long non-coding RNAs as a determinant of cancer drug resistance: Towards the overcoming of chemoresistance via modulation of IncRNAs. Drug Resistance Updates, 50, 100683). Например, днРНК MALAT1 в значительной степени ассоциирована с метастазированием рака. Было также показано, что нокдаун MALAT1 в клетках глиобластомы увеличивал чувствительность к темозоломиду (Cai et al. (2018). Long noncoding RNA MALAT 1 knockdown reverses chemoresistance to temozolomide via promoting micro RNA-101 in glioblastoma. Cancer Medicine, 7(4), 1404-1415). MALAT1 содержит на 3'-конце тройную спиральную структуру, которая защищает эту РНК от деградации экзонуклеазой и способствует ее ядерной аккумуляции. Была обнаружена малая молекула на основе дифенилфурана, которая специфически нацелена на структурный элемент тройной спирали в MALAT1 (Donlic et al. (2018). Discovery of small molecule ligands for MALAT1 by tuning an RNA-binding scaffold. Angewandte Chemie, 130(40), 13426-13431). Однако для проведения клинических исследований еще предстоит выяснить, приводит ли связывание этой молекулы к эффективной дестабилизации MALAT1.
Терапия, нацеленная на днРНК стала предметом исследований только в последнее десятилетие, и до сих пор ни один препарат, нацеленный на днРНК, не вышел в клиническую разработку. В будущем ожидается, что днРНК расширят круг мишеней для РНК-интерференции и методов CRISPR Cas, а тканеспецифичные типы днРНК, например, энхансерные РНК (эРНК), позволят использовать совершенно новые терапевтические подходы. Энхансерные РНК транскрибируются с активных энхансерных областей, а поскольку активность энхансеров часто является тканеспецифичной, то и экспрессия эРНК может отличаться в зависимости от типа клеток. Например, нашей группой была найдена эРНК, которая влияет на клеточный рост и устойчивость к химиотерапии клеток лимфомы Беркитта, в то время как на здоровые CD19 клетки схожего эффекта обнаружено не было (Stasevich et al (2022). Enhancer RNA AL928768.3 from the IGH locus regulates MYC expression and controls the proliferation and chemoresistance of Burkitt lymphoma cells with IGH/MYC translocation. International Journal of Molecular Sciences, 23(9), 4624).
Таким образом, существующие в настоящее время подходы к лечению глиобластомы требуют улучшения. Многочисленные исследования в данной области подтверждают важность и актуальность работ, направленных на увеличение медианы выживаемости пациентов с глиобластомой, в частности, решающих проблему устойчивости к классической терапии темозоломидом.
Предлагаемый способ снижения пролиферации глиобластомы, заключающийся в комбинации темозоломида и подавление экспрессии эРНК с помощью РНК-интерференции, является многообещающим подходом для терапии и может помочь в решении актуальной задачи.
Раскрытие сущности изобретения
Техническим результатом изобретения является подавление пролиферации клеток глиобластомы, достигаемое посредством преодоления устойчивости к классическому химиотерапевтическому агенту темозоломиду.
Технический результат достигается за счет использования комбинации классической химиотерапии темозоломидом и терапии РНК-препаратами (метод РНК-интерференции).
Предлагаемый способ является новым и не описан в патентной и научно-технической литературе.
Краткое описание фигур и таблиц
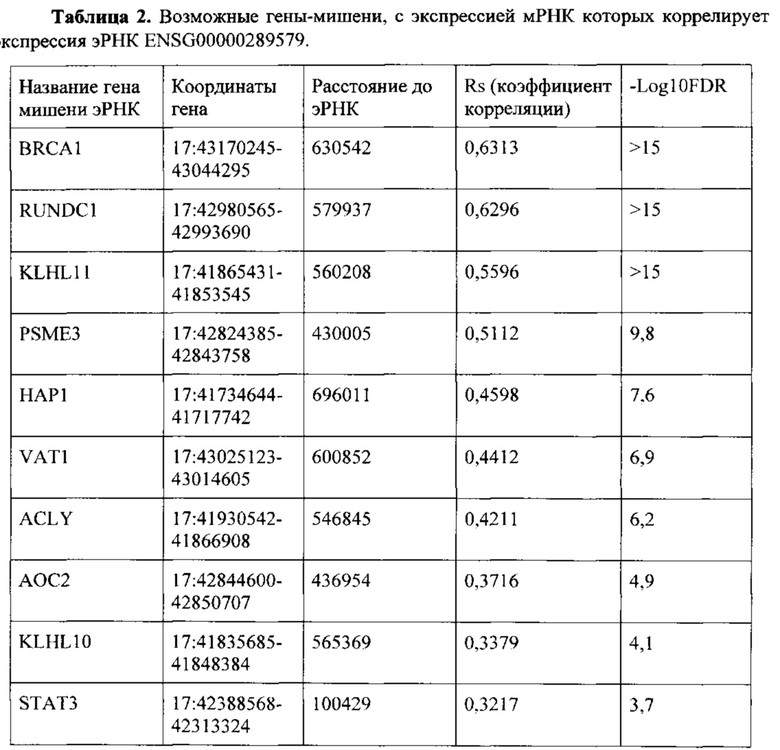
Фиг. 1. Нокдаун эРНК ENSG00000289579 в клеточной линии глиобластомы DBTRG-05MG. Представлена измеренная методом количественного ПЦР относительная экспрессия эРНК ENSG00000289579 при доставке в клетки малых интерферирующих РНК (siRNA). Темозоломид (TMZ) использовался в концентрации 20 мкг/мл, в качестве растворителя - диметилсульфоксид (DMSO). Представлен результат 6 независимых биологических повторов. Для нормировки использовался ген домашнего хозяйства GAPDH. * р-значение менее 0,05 (критерий Манна-Уитни).
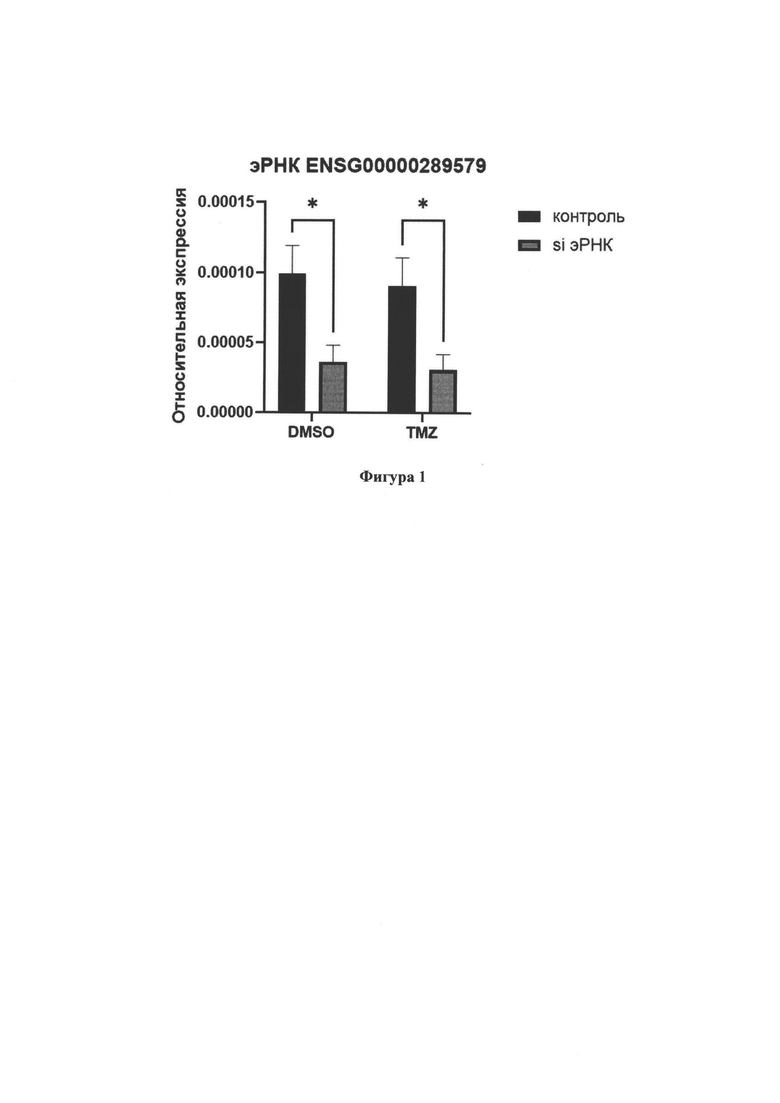
Фиг. 2. Влияние на клеточный рост нокдауна эРНК ENSG00000289579 в клеточной линии глиобластомы DBTRG-05MG. Представлена измеренная с помощью прибора xCELLigence RTCA DP кривая роста клеточной линии DBTRG-05MG при добавлении специфичных малых интерферирующих РНК (siRNA). Темозоломид (TMZ) использовался в концентрации 20 мкг/мл, в качестве растворителя - диметилсульфоксид (DMSO), поскольку темозоломид хранится растворенным в DMSO. Представлен результат 6 независимых биологических повторов.
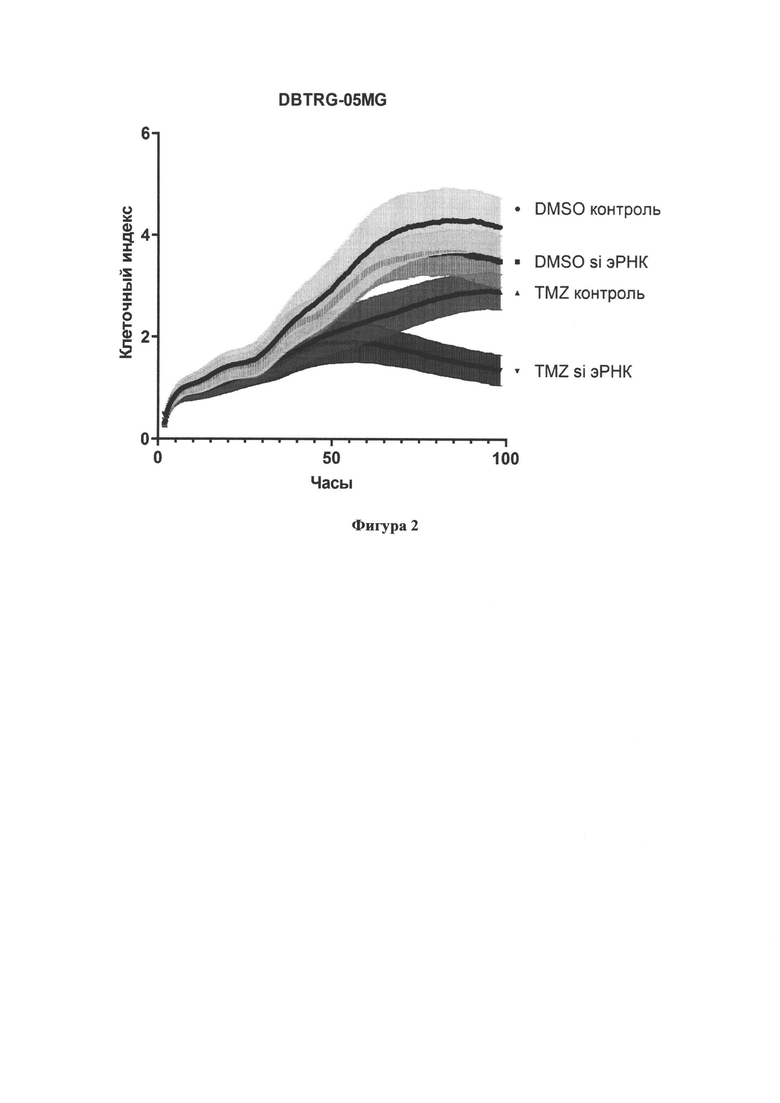
Фиг. 3. Влияние на клеточный рост нокдауна эРНК ENSG00000289579 в клеточной линии глиобластомы DBTRG-05MG через 96 часов. Представлен измеренный с помощью прибора xCELLigence RTCA DP клеточный индекс клеток DBTRG-05MG при добавлении специфичных малых интерферирующих РНК (siRNA) через 96 часов после начала эксперимента. Темозоломид (TMZ) использовался в концентрации 20 мкг/мл, в качестве растворителя - диметилсульфоксид (DMSO), поскольку темозоломид хранится растворенным в DMSO. Представлен результат 6 независимых биологических повторов. ** р-значение менее 0,01 (критерий Манна-Уитни).
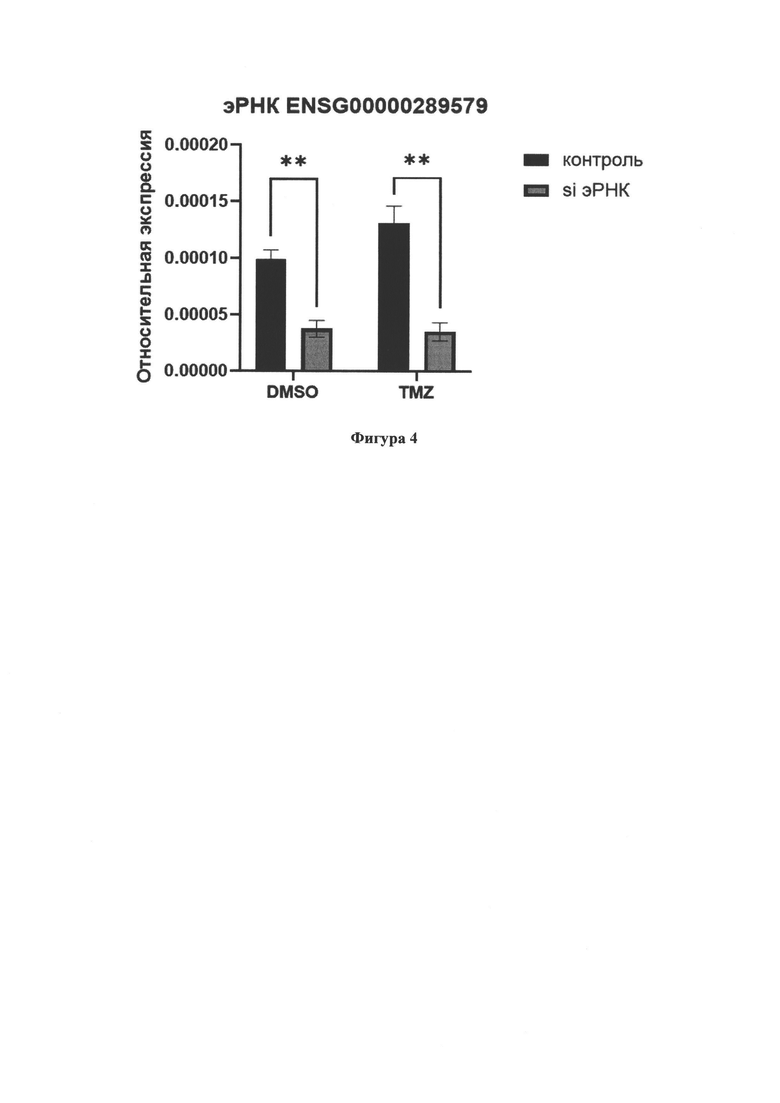
Фиг. 4. Нокдаун эРНК ENSG00000289579 в клеточной линии глиобластомы T98G. Представлена измеренная методом количественного ПЦР относительная экспрессия эРНК ENSG00000289579 в случае доставки в клетки малых интерферирующих РНК (siRNA). Темозоломид (TMZ) использовался в концентрации 20 мкг/мл, в качестве растворителя - диметилсульфоксид (DMSO), поскольку темозоломид хранится растворенным в DMSO. Представлен результат 4 независимых биологических повторов. Для нормировки использовался ген домашнего хозяйства GAPDH. ** р-значение менее 0,01 (критерий Манна-Уитни).
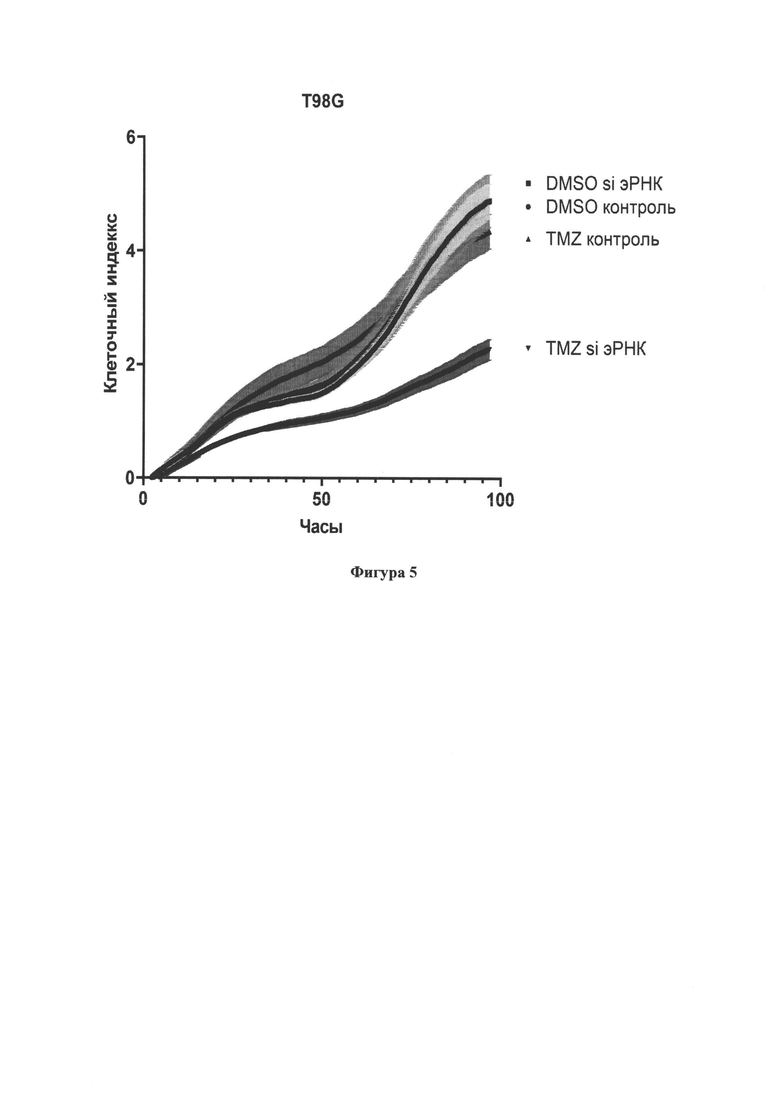
Фиг. 5. Влияние на клеточный рост нокдауна эРНК ENSG00000289579 в клеточной линии глиобластомы T98G. Представлена кривая роста клеточной линии T98G при добавлении специфичных малых интерферирующих РНК (siRNA). Темозоломид (TMZ) использовался в концентрации 20 мкг/мл, в качестве растворителя - диметилсульфоксид (DMSO), поскольку темозоломид хранится растворенным в DMSO. Представлен результат 4 независимых биологических повторов.
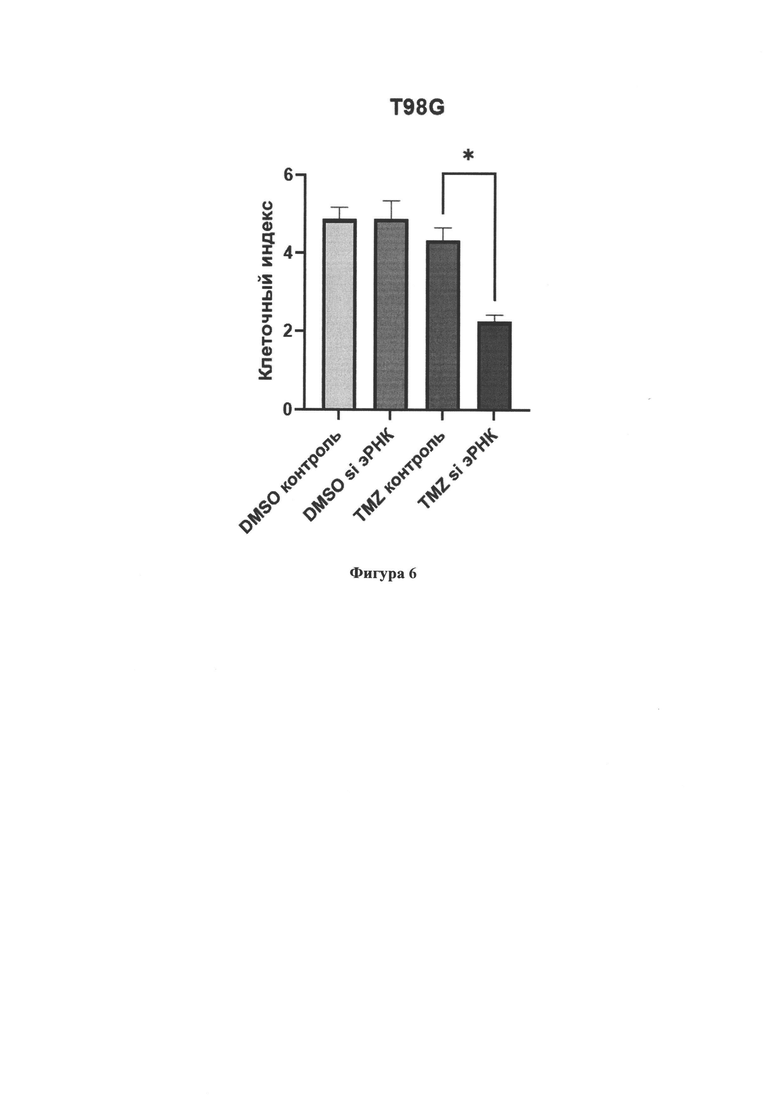
Фиг. 6. Влияние на клеточный рост нокдауна эРНК ENSG00000289579 в клеточной линии глиобластомы DBTRG-05MG через 96 часов. Представлен измеренный с помощью прибора xCELLigence RTCA DP клеточный индекс клеток DBTRG-05MG при добавлении специфичных малых интерферирующих РНК (siRNA) через 96 часов. Темозоломид (TMZ) использовался в концентрации 20 мкг/мл, в качестве растворителя - диметилсульфоксид (DMSO), поскольку темозоломид хранится растворенным в DMSO. Представлен результат 4 независимых биологических повторов. * р-значение менее 0,05 (критерий Манна-Уитни).
Табл. 1. Последовательность малых интерферирующих РНК. В последовательностях SEQ ID NO: 1-4 два последних остатка представляют собой дезокситимидин (Т).
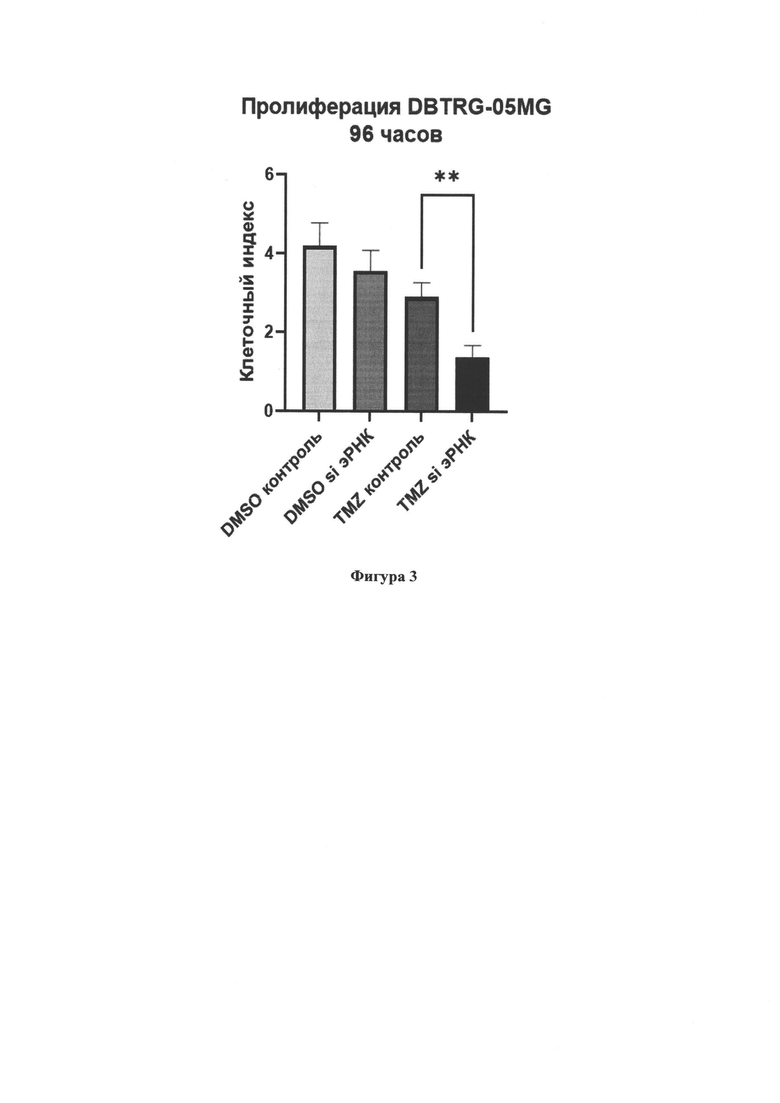
Табл. 2. Возможные гены-мишени, с экспрессией мРНК которых коррелирует экспрессия эРНК ENSG00000289579. Представлен список генов мишеней эРНК ENSG00000289579, координаты генов (сборка генома hg38), расстояние до эРНК, коэффициент корреляции эРНК ENSG00000289579 с конкретным геном и отрицательный десятичный логарифм False discovery rate.
Осуществление изобретения
Описанное изобретение направлено на разработку нового подхода в подавлении пролиферации клеток глиобластомы, в частности в преодолении резистентности клеток глиобластомы к темозоломиду.
Предлагаемый подход представляет собой улучшение и исправление широко используемого подхода для лечения глиобластомы. Предлагаемая комбинация двух компонентов, а именно: классической химиотерапии темозоломидом с использованием РНК-интерференции для подавления экспрессии эРНК ENSG00000289579, что обеспечивает значительное снижение пролиферации опухолевых клеток и уход клеток глиобластомы от резистентности к темозоломиду.
Для подавления экспрессии эРНК ENSG00000289579 используется малая интерферирующая РНК, которая содержит смысловую цепь, представленную последовательностью SEQ ID NO: 1 и антисмысловую цепь - SEQ ID NO: 2, поставляемые в виде лиофилизатов (ДНК-Синтез), к каждой последовательности РНК были добавлены 2 нуклеотида dT для стабилизации (Табл. 1). Лиофилизированные олигонуклеотиды растворяют в буфере 10 мМ Tris-HC1, 2,5 мМ MgCl2, 0,1 мМ СаCl2 (рН 7.5) при температуре 25°С или в специальном буфере siRNA Buffer, содержащем хлорид калия и хлорид магния, в концентрации 100-500 пМоль. Далее одинаковое молярное количество смысловой и антисмысловой РНК смешиваются, ставятся в водяную баню при температуре 95°С и затем остужаются не быстрее 1 градуса в минуту. Растворенная малая интерферирующая РНК хранится при -20°С и ниже.
Доставка малой интерферирующей РНК в клетки осуществляется различными способами, например такими как липофекция (Lipofectamine™ RNAiMAX, GenJect™-40 или другой липофекционный агент), электропорация (система трансфекции Neon, Lonza и другие).
Энхансерная РНК ENSG00000289579 имеет ряд потенциальных мишеней, воздействие на которые может приводить к снижению пролиферации клеток глиобластомы (Табл. 2). Список потенциальных генов мишеней эРНК ENSG00000289579 (ENSR00000094395) был получен с помощью базы данных энхансерных РНК в раке eRic (https://hanlab.uth.edu/eRic/).
Далее настоящее изобретение детально проиллюстрировано с использованием конкретного примера, представляющего возможное воплощения изобретения. В то же время изобретение не ограничивается описанными воплощениями, а включает любые альтернативные варианты и модификации, применение которых возможно с учетом сущности изобретения.
Пример 1. Увеличение чувствительности клеточных линий DBTRG-05MG и T98G к темозоломиду с помощью нокдауна эРНК ENSG00000289579 доставкой малых интерферирующих РНК липофекцией
Клеточные линии и трансфекция. Клеточные линии глиобластомы DBTRG-05MG и T98G ведут в среде DMEM с добавлением 10% эмбриональной бычьей сыворотки (Corning), 50 Ед/мл пенициллина и 50 мкг/мл стрептомицина (ПанЭко), 1 × аминокислоты заменимые (ПанЭко), 10 мМ HEPES (ПанЭко). Трансфекцию малых интерферирующих РНК (siRNA) производят с помощью трансфекционного реагента Lipofectamine RNAiMAX (Thermo FS) как указано в протоколе. В качестве контроля используют малые инферирующие РНК с рандомизированной последовательностью, не имеющей мишеней в человеческих клетках, представленных последовательностями SEQ ID NO: 3 и SEQ ID NO: 4 (Табл. 1).
Измерение клеточной пролиферации и химиорезистентности. Для оценки клеточного роста и химиорезистентности сажают 1,5*104 клеток на лунку клеточного анализатора xCelligence. Каждая точка измеряется в двух технических повторах. Через час после посадки клеток добавляют темозоломид (TMZ) в концентрации 20 мкг/мл. В качестве контроля используют диметилсульфоксид (DMSO), поскольку темозоломид был растворен в диметилсульфоксиде. Измерение клеточной пролиферации (клеточного индекса) производят каждые 15 минут в течение 96 часов.
Выделение тотальной РНК и постановка ПЦР в реальном времени. Через 24 часа после начала эксперимента из клеток глиобластомы выделяют тотальную РНК (ExtractRNA, Евроген), затем образцы обрабатывают ДНКазой (Thermo Scientific) для избавления от возможных примесей геномной ДНК и производят синтез первой цепи ДНК с использование обратной транскриптазы MMLV (Евроген) в присутствии рандомных и олигоdТ праймеров в соотношении 1:1 в соответствии с протоколом производителя. Экспрессию эРНК ENSG00000289579 оценивают метод ПЦР в реальном времени с использованием специфичных праймеров SEQ ID NO: 5 и SEQ ID NO: 6. Для нормировки используют ген домашнего хозяйства GAPDH (последовательности SEQ ID NO: 7 и SEQ ID NO: 8).
По базам данных чувствительности разных клеточных линий к химотерапии Genomics of Drug Sensitivity in Cancer (GDSC1, GDSC2) клеточные линии глиобластомы DBTRG-05MG и T98G являются устойчивыми клеточными клетками к темозоломиду.
Был произведен нокдаун в клеточной линии DBTRG-05MG. Было подтверждено, что экспрессия эРНК ENSG00000289579 снижается при добавлении малых интерферирующих РНК (Фиг. 1). Наблюдалось сильное снижение пролиферации в комбинации темозоломида и малых интерферирующих РНК к эРНК ENSG00000289579 (Фиг. 2, 3).
Нокдаун в клеточной линии T98G приводил также к значимому снижению экспрессии эРНК ENSG00000289579 (Фиг. 4). По результатам наблюдалось сильное снижение пролиферации клеточной линии T98G при использовании комбинации темозоломида и малых интерферирующих РНК к эРНК ENSG00000289579 по сравнению с контрольными образцами (Фиг. 5, 6).

--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="ru"
dtdVersion="V1_3" fileName="СПОСОБ СНИЖЕНИЯ УСТОЙЧИВОСТИ КЛЕТОК
ГЛИОБЛАСТОМЫ К ТЕМОЗОЛОМИДУ.xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2023-09-23">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText></ApplicationNumberText>
<FilingDate>2023-09-14</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>-</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное учреждение науки Институт молекулярной биологии им. В.А.
Энгельгардта Российской академии наук (ИМБ РАН)</ApplicantName>
<ApplicantNameLatin>Engelhardt Institute of Molecular Biology,
Russian Academy of Sciences</ApplicantNameLatin>
<InventionTitle languageCode="ru">СПОСОБ СНИЖЕНИЯ УСТОЙЧИВОСТИ
КЛЕТОК ГЛИОБЛАСТОМЫ К ТЕМОЗОЛОМИДУ</InventionTitle>
<SequenceTotalQuantity>8</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q7">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q8">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>sense RNA</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gccagatctcgagtttagacatt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q11">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>antisense RNA</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgtctaaactcgagatctggctt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q12">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>direct primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtgtaccaacgactcaatgttt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q13">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>reverse primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acattgagtcgttggtacacctt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q15">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q16">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>specific reverse
prime</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagagcctcctacccagacc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q19">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>specific reverse
primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccaagcacacatagctcctca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q21">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q22">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>GAPDH direct primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caaggtcatccatgacaactttg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q25">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>GAPDH reverse
primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggccatccacagtcttctgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| КОМБИНИРОВАННАЯ ТЕРАПИЯ ГЕПАТОЦЕЛЛЮЛЯРНОЙ КАРЦИНОМЫ НА ОСНОВЕ СОРАФЕНИБА | 2022 |
|
RU2800071C1 |
| Двухцепочечная РНК, способная снижать экспрессию мутантного аллеля с.607GA гена GNAO1 человека | 2022 |
|
RU2816137C1 |
| ПРИМЕНЕНИЕ АНТИТЕЛА ПРОТИВ О-АЦЕТИЛИРОВАННОГО ГАНГЛИОЗИДА GD2 ДЛЯ УЛУЧШЕНИЯ ТЕРАПЕВТИЧЕСКОГО ПОТЕНЦИАЛА ЛЕКАРСТВЕННЫХ СРЕДСТВ | 2017 |
|
RU2771173C2 |
| ПРЕПАРАТЫ, ОСУЩЕСТВЛЯЮЩИЕ РНК-ИНТЕРФЕРЕНЦИЮ, ДЛЯ МОДУЛЯЦИИ ГЕНА GST-PI | 2015 |
|
RU2719185C2 |
| ЛИНИИ СКОНСТРУИРОВАННЫХ КЛЕТОК-ПРОДУЦЕНТОВ И СПОСОБЫ ИХ ПОЛУЧЕНИЯ И ПРИМЕНЕНИЯ | 2020 |
|
RU2823068C2 |
| ОЛИГОНУКЛЕОТИДНЫЕ ПОСЛЕДОВАТЕЛЬНОСТИ, НАЦЕЛЕННЫЕ НА ТРАНСКРИПЦИОННЫЙ ФАКТОР TSC22D4, ДЛЯ ЛЕЧЕНИЯ РЕЗИСТЕНТНОСТИ К ИНСУЛИНУ | 2016 |
|
RU2723091C2 |
| Оптимизированный штамм полиовируса 3 типа для терапии солидных опухолей | 2023 |
|
RU2836222C1 |
| Композиция на основе лития цитрата и мелатонина с антиглиобластомным действием | 2022 |
|
RU2787883C1 |
| Способ анализа соматических мутаций в генах IDH1 и IDH2 с использованием LNA-блокирующей ПЦР и гибридизации с биологическим микрочипом | 2024 |
|
RU2839291C1 |
| ПРИМЕНЕНИЕ АЛЬФАВИРУСА В ПОЛУЧЕНИИ ПРОТИВООПУХОЛЕВЫХ ЛЕКАРСТВЕННЫХ СРЕДСТВ | 2015 |
|
RU2693938C2 |
Изобретение относится к области биотехнологии, в частности к способу снижения пролиферации клеток глиобластомы. Указанный способ включает доставку в клетки глиобластомы малых интерферирующих РНК, представленных последовательностями SEQ ID NO: 1 и SEQ ID NO: 2, в комбинации с химиотерапией темозоломидом TMZ. Изобретение обеспечивает подавление пролиферации клеток глиобластомы, что достигается посредством преодоления устойчивости к классическому химиотерапевтическому агенту темозоломиду. 2 з.п. ф-лы, 6 ил., 2 табл., 1 пр.
1. Способ снижения пролиферации клеток глиобластомы, включающий доставку в клетки глиобластомы малых интерферирующих РНК, представленных последовательностями SEQ ID NO:1 и SEQ ID NO:2, в комбинации с химиотерапией темозоломидом TMZ.
2. Способ по п. 1, характеризующийся тем, что с помощью малых интерферирующих РНК SEQ ID NO: 1 и SEQ ID NO: 2 проводят нокдаун эРНК ENSG00000289579.
3. Способ по п. 2, характеризующийся тем, что снижение экспрессии эРНК ENSG00000289579 приводит к увеличению чувствительности клеток глиобластом к темозоломиду.
| DING, JIE et al | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Разборный с внутренней печью кипятильник | 1922 |
|
SU9A1 |
| Приспособление для удержания оборвавшейся подъемной цепи или ленты элеватора | 1925 |
|
SU2428A1 |
| SHAN, X | |||
| et al | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
Авторы
Даты
2024-11-07—Публикация
2023-10-25—Подача