Область техники
Настоящее изобретение относится к области медицины, а именно к онкологии, и может быть использовано для лечения онкологических заболеваний.
Уровень техники
Диагностика гепатоцеллюлярной карциномы (ГЦК) в основном основывается на неинвазивных критериях, что значительно усложняет раннее распознавание заболевания. Резекция и трансплантация печени до сих пор остаются основными методами лечения ГЦК (J. Bruix et al. Management of hepatocellular carcinoma. 2005). Однако за последние 5 лет достигнут значительный прогресс в разработке системных методов терапии, основанных на использовании ингибиторов контрольных точек иммунного ответа (антитела к PD-1, PD-L1, CTLA-4, например, Ниволумаб, Пембролизумаб), внутриклеточных киназ (серин/треонинкиназы c-CRAF, BRAF) и тирозинкиназ (Сорафениб, Сунитиниб, Орантиниб, Бриваниб, Линифаниб, Регорафениб), моноклональных антител, которые начинают замещать традиционные методы лечения ГЦК (J. Llovet et al. Molecular therapies and precision medicine for hepatocellular carcinoma. 2018). Сорафениб и ленватиниб являются наиболее эффективными однокомпонентными терапевтическими препаратами (M. Kudo et al. Lenvatinib versus sorafenib in first-line treatment of patients with unresectable hepatocellular carcinoma: a randomised phase 3 non-inferiority trial. 2018).
Сорафениб (BAY 43-9006, Nexavar®; Bayer Healthcare Pharmaceuticals Inc.; Леверкузен, Германия) был первым системным препаратом, одобренным управлением по санитарному контролю за качеством пищевых продуктов и медикаментов США (FDA) для лечения неоперабельного рака печени (https://www.accessdata.fda.gov/drugsatfda_docs/label/2010/021923s008s009lbl.pdf).
Сорафениб действует как мультикиназный ингибитор белков, регулирующих клеточный цикл, и таким образом блокирует пролиферацию опухолевых клеток. Однако у многих пациентов появляется резистентность к препарату из-за генетической гетерогенности ГЦК. Также стоит учитывать побочные эффекты от приема сорафениба, такие как сыпь, диарея, высокое кровяное давление и синдром "рука-нога", что ограничивает терапевтическое окно для применения препарата. В связи с гетерогенной природой ГЦК перспективно сочетать сорафениб с другими противоопухолевыми агентами. В настоящее время проводятся исследования, изучающие комбинацию сорафениба и трансартериальной химиоэмболизации (TACE), однако эффективность этого лечения еще не продемонстрирована (R. Lencioni. Sorafenib or placebo plus TACE with doxorubicin-eluting beads for intermediate stage HCC: The SPACE trial. 2016). На стадии клинических испытаний находится адъювантное лечение сорафенибом в комбинации с ECOG1208 (NCT01004978), TACE-2 (NCT01324076) и TACTICS (NCT01217034) (Y. Zhu et al. New knowledge of the mechanisms of sorafenib resistance in liver cancer. 2017). Как научные, так и клинические исследования показывают, что сочетание сорафениба с другими лекарственными средствами оказывает благоприятный эффект и обеспечивает новый подход к лечению.
Однако существующие терапевтические агенты и подходы к лечению ГЦК обладают рядом недостатков, включая резистентность, ограниченное терапевтическое окно, побочные эффекты и невысокую медиану выживаемости пациентов, что определяет необходимость разработки новых комбинаторных препаратов и способов терапии.
Одним из новых терапевтических подходов к лечению различных заболеваний является терапия на основе РНК-интерференции, процесса селективного подавления экспрессии генов за счет деградации мРНК, опосредованного малыми интерферирующими РНК (миРНК, siRNA). Первый препарат на основе миРНК - Патисиран (A. Mullard. FDA approves landmark RNAi drug. 2018) был одобрен для применения в клинике в 2018 году. А в 2019 году другой препарат на основе миРНК, Гивосиран (Givlaari, Alnylam Pharmaceuticals), получил одобрение управления по контролю за продуктами и лекарствами (FDA) для лечения острой порфирии печени (Givlaari. https://www.givlaari.com. 2020), а многие другие находятся в разработке. Традиционные подходы к терапии с использованием низкомолекулярных препаратов основаны на их взаимодействии с активными участками белков таким образом, чтобы подавлять или изменять их функцию. Однако только 1,5 % человеческого генома кодирует белки (I. Ezkurdia et al. Multiple evidence strands suggest that there may be as few as 19 000 human protein-coding genes. 2014) и всего 10-14 % белков имеют активные сайты связывания, которые являются потенциальными селективными мишенями для малых молекул (A. Hopkins, C. Groom. The druggable genome. 2002). Использование рекомбинантных белков в качестве лекарственных агентов решило часть проблем и расширило подходы к терапии. Однако рекомбинантные белки имеют свои ограничения в применении, связанные с их размером и стабильностью и необходимостью наличия посттрансляционных модификаций, которые усложняют процесс синтеза (Z. Antosova et al. Therapeutic application of peptides and proteins: parenteral forever? 2009). В тоже время подходы, основанные на применении нуклеиновых кислот, не попадают под многие из этих ограничений, поскольку они регулируют экспрессию мишени до этапа трансляции. РНК-интерференция представляет собой перспективный подход для лечения онкологических заболеваний, например, путём выключения генов, характеризующихся повышенной экспрессией в раковых клетках, либо генов, принимающих участие в делении клетки. Кроме того, на сегодняшний день важным направлением исследований в области РНК-интерференции для клинического применения является разработка способов безопасной и эффективной доставки малых РНК (Haussecker D. Current issues of RNAi therapeutics delivery and development // Journal of Controlled Release. 2014. V.195, pp.49-54; Mansoori B. et al. RNA Interference and its role in cancer therapy // Adv. Pharm. Bull. 2014. V4(4), pp.313-321).
Все известные на сегодняшний день системы доставки миРНК можно разделить на два типа: вирусные и невирусные векторы. Применение вирусных систем имеет ряд ограничений, таких как высокая стоимость и иммунный ответ (R. Tomar et al. Use of adeno-associated viral vector for delivery of small interfering RNA. 2003). Липидные наночастицы (ЛНЧ) относятся к невирусным системам доставки миРНК и обладают рядом преимуществ: низкая токсичность, высокая эффективность интернализации, низкая стоимость, легкое масштабирование синтеза (Y. Xin et al. Nano-based delivery of RNAi in cancer therapy. 2017). Инкапсуляция терапевтической миРНК в липидные наночастицы для адресной доставки была использована в лекарственном средстве Патисиран для лечения транстиретин-опосредованной наследственной амилоидной полинейропатии, одобренного управлением по контролю за продуктами и лекарствами (FDA) в 2018 году (J. Kulkarni et al. Lipid nanoparticle technology for clinical translation of siRNA therapeutics. 2019; D. Adams et al. Patisiran, an RNAi therapeutic, for hereditary transthyretin amyloidosis. 2018).
Одной из перспективных мишеней для терапии онкозаболеваний является ганкирин (PSMD10). Изначально белок был идентифицирован как компонент протеасомы 26S (S. Dawson et al. Gankyrin is an ankyrin-repeat oncoprotein that interacts with CDK4 kinase and the S6 ATPase of the 26 S proteasome. 2002). Ганкирин участвует в различных биологических процессах, включая клеточный рост, пролиферацию, инвазию и регенерацию печени (Y. Shan et al. The role of p28GANK in rat oval cells activation and proliferation. 2006). За счет взаимодействия с белком ретинобластомы (Rb) и циклин-зависимой киназы 4 (CDK4) ганкирин способствует фосфорилированию и инактивации Rb, что в свою очередь приводит к активации фактора транскрипции E2F и движению по клеточному циклу (S. Dawson et al. Gankyrin: a new oncoprotein and regulator of pRb and p53. 2006; R. Mayer et al. Gankyrin, the 26 S proteasome, the cell cycle and cancer. 2006). Также ганкирин способствует убиквитинилированию и деградации p53 за счет связывания с белком комплекса PRC2-Mdm2 (H. Higashitsuji et al. The oncoprotein gankyrin binds to MDM2/HDM2, enhancing ubiquitylation and degradation of p53.2005). Ганкирин участвует в Ras-зависимом канцерогенезе и участвует в регуляции Act/mTOR сигнального пути (J. Man et al. Gankyrin plays an essential role in Ras-induced tumorigenesis through regulation of the RhoA/ROCK pathway in mammalian cells. 2010). Не так давно было показано участие ганкирина в регуляции PI3K/AKT, IL-6/STAT3, IL-1β/IRAK-1 и β-катенинового сигнальных путей (J. Zhang et al. Gankyrin plays an essential role in estrogen-driven and GPR30-mediated endometrial carcinoma cell proliferation via the PTEN/PI3K/AKT signaling pathway. 2013; T. Zheng et al. Gankyrin promotes tumor growth and metastasis through activation of IL-6/STAT3 signaling in human cholangiocarcinoma. 2014; L. Dong et al. The oncoprotein p28GANK establishes a positive feedback loop in beta-catenin signaling. 2011; B. Su et al. Interleukin-1β/Iinterleukin-1 receptor-associated kinase 1 inflammatory signaling contributes to persistent Gankyrin activation during hepatocarcinogenesis. 2015). Ганкирин участвует в регуляции NF-κB/RelA на транскрипционном уровне, связываясь с RelA и модулируя эффективность его ацилирования (H. Higashitsuji et al. The oncoprotein gankyrin interacts with RelA and suppresses NF-kappaB activity. 2007).
Многочисленные данные подтверждают онкогенные функции ганкирина в развитии ГЦК и других видах онкозаболеваний: глиоме, липосаркоме и раке легких, пищевода, желудка, толстой кишки, поджелудочной железы, шейки матки, яичников, эндометрия и молочной железы (J. Fu et al. p28GANK overexpression accelerates hepatocellular carcinoma invasiveness and metastasis via phosphoinositol 3-kinase/AKT/hypoxia-inducible factor-1α pathways. 2011; Y. Yang et al. Up-regulated oncoprotein P28GANK correlates with proliferation and poor prognosis of human glioma. 2012; W. Wang et al. Clinicopathologic features and prognostic implications of Gankyrin protein expression in non-small cell lung cancer. 2015; Y. Liu et al. Association between functional PSMD10 Rs111638916 variant regulated by MiR-505 and gastric cancer risk in a Chinese population. 2015; S. Tang et al. Overexpression of a novel gene gankyrin correlates with the malignant phenotype of colorectal cancer. 2010; Y. Meng et al. Gankyrin promotes the proliferation of human pancreatic cancer.2010).
Стоит отметить, что экспрессия ганкирина практически отсутствует в нормальных гепатоцитах, слабо выражена при циррозе и паракарциноме печени и наиболее явно представлена в печени при ГЦК. Также показано, что ганкирин может способствовать прогрессированию и метастазированию ГЦК, что делает данный белок онкомаркером и перспективной мишенью для терапии (Y. Fu et al. Overexpression of p28/gankyrin in human hepatocellular carcinoma and its clinical significance. 2002). Не так давно было показано, что ганкирин способствует активации аутофагии в клетках в ответ на химиотерапию ГЦК (T. Luo et al. PSMD10/gankyrin induces autophagy to promote tumor progression through cytoplasmic interaction with ATG7 and nuclear transactivation of ATG7 expression. 2016).
Для регулирования уровня ганкирина используется низкомолекулярный ингибиритор гистоновой деацетилазы - панобиностат (X. Song et al. LBH589 inhibits proliferation and metastasis of hepatocellular carcinoma via inhibition of gankyrin/STAT3/Akt pathway. 2013). Также было показано, что арктигенин (компонент лопуха) снижает уровень ганкирина в гепатоцитах, что приводит к остановке клеточного роста (Y. Sun et al. Arctigenin inhibits liver cancer tumorigenesis by inhibiting Gankyrin expression via C/EBPα and PPARα. 2018).
Исследования на животных показали, что снижение уровня ганкирина в непаренхимальных клетках подавляет активность транскрипционного фактора Stat3, синтез интерлейкина IL-6 и маркеров раковых стволовых клеток (Bmi1, EpCAM), тем самым снижая метастазирование. Более того, исследование показало, что количество ганкирина в опухоли обратно пропорционально медиане выживаемости пациентов с ГЦК, получавших лечение сорафенибом (T. Sakurai et al. Gankyrin induces STAT3 activation in tumor microenvironment and sorafenib resistance in hepatocellular carcinoma. 2017).
Несмотря на многочисленные исследования в поиске различных подходов к лечению опухолевых заболеваний печени, разработка эффективного препарата для терапии пациентов с такими заболеваниями, способного увеличить медиану выживаемости пациентов, до сих пор является очень актуальной.
Раскрытие изобретения
Задачей предлагаемого изобретения является разработка гепатотропных липидных наночастиц и нового лекарственного препарата на основе этих наночастиц, эффективного для лечения опухолевых заболеваний печени, а также разработка метода лечения указанных заболеваний с использованием такого препарата.
Предлагаемый подход принципиально отличается от уже существующих средств и способов терапии опухолевых заболеваний печени, поскольку представляет собой комбинацию двух подходов - классической химиотерапии сорафенибом с принципиально новым типом терапии с использованием РНК-интерференции, обеспечивающим подавление экспрессии ганкирина, регулирующего пролиферацию опухолевых клеток.
Поставленная задача решается путем разработки нового лекарственного препарата, включающего миРНК, ингибирующие экспрессию ганкирина, и сорафениб, инкапсулированные совместно в липидные наночастицы. Причем структура миРНК, их модификация, а также структура наночастиц разработаны таким образом, чтобы обеспечить максимальную эффективность и биодоступность предлагаемого препарата.
Более конкретно, решение поставленной задачи достигается путем разработки липидных наночастиц (ЛНЧ) для лечения опухолевых заболеваний печени, содержащих:
(1) малую интерферирующую РНК (миРНК), способную ингибировать экспрессию ганкирина, где миРНК представляет собой дуплекс, содержащий
а) смысловую цепь GccGGGAuGAGAuuGuAAATsT (SEQ ID NO: 1) и антисмысловую цепь UUuAcAAUCUcAUCCCGGCTsT (SEQ ID NO: 2) или
б) смысловую цепь cAAucAAAAuGGcuGuAcuTsT (SEQ ID NO: 5) и антисмысловую цепь AGuAcAGCcAUUUUGAUUGTsT (SEQ ID NO: 6),
где нуклеотиды, обозначенные строчными буквами, представляют собой 2'-O-метилрибонуклеотиды; s представляет собой тиофосфатную связь,
и (2) 4-[4-({[4-хлоро-3-(трифторометил)фенил]карбамоил}амино)фенокси]-N-метил-пиридин-2-карбоксамид (сорафениб) или его фармацевтически приемлемую соль,
инкапсулированные совместно в липидные наночастицы (ЛНЧ), изготовленные на основе липидоида C12-200.
Более конкретно, липидные наночастицы содержат 45-50% липидоида С12-200, 10-20% дистеароилфосфатидилхолина, 35-38,5% холестерина и 0,4-1,5% полиэтиленгликоля PEG2000-C14. В частных вариантах воплощения изобретения липидные наночастицы содержат 50% липидоида С12-200, 10% дистеароилфосфатидилхолина, 38,5% холестерина и 1,5% полиэтиленгликоля PEG2000-C14.
В некоторых вариантах изобретения содержание компонента (1) в липидных наночастицах по отношению к компоненту (2) составляет 5-8 мол.%.
Решение поставленной задачи также достигается путем разработки фармацевтической композиции для лечения опухолевых заболеваний печени у пациента, в форме гепатотропных липидных наночастиц для парентерального (в предпочтительных вариантах - внутривенного) введения, содержащей в качестве терапевтически активного ингредиента липидные наночастицы, определенные выше, в эффективном количестве и фармацевтически приемлемый носитель, представляющий собой воду.
В некоторых частных вариантах вода представляет собой деионизованную воду или воду для инъекций.
Более конкретно, фармацевтическая композиция по изобретению представляет собой композицию, содержащую в качестве терапевтически активного ингредиента:
(1) малую интерферирующую РНК (миРНК), способную ингибировать экспрессию ганкирина, где миРНК представляет собой дуплекс, содержащий
а) смысловую цепь, имеющую последовательность SEQ ID NO: 1 и антисмысловую цепь, имеющую последовательность SEQ ID NO: 2 или
б) смысловую цепь, имеющую последовательность SEQ ID NO: 5 и антисмысловую цепь, имеющую последовательность SEQ ID NO: 6,
и (2) 4-[4-({[4-хлоро-3-(трифторометил)фенил]карбамоил}амино)фенокси]-N-метил-пиридин-2-карбоксамид или его фармацевтически приемлемую соль,
в терапевтически эффективных количествах, инкапсулированные совместно в липидные наночастицы (ЛНЧ), изготовленные на основе липидоида C12-200.
Согласно изобретению, липидные наночастицы в фармацевтической композиции находятся в водной фазе.
В некоторых вариантах изобретения фармацевтическая композиция приготовлена в виде готовой липосомной (липидной) формы.
Решение поставленной задачи также достигается путем разработки способа получения липидных наночастиц по изобретению, включающего следующие стадии:
получение первого раствора, представляющего собой водную фазу, содержащую 0,1-0,4 мг/мл миРНК (имеющей смысловую цепь, характеризующуюся последовательностью SEQ ID NO: 1 и антисмысловую цепь, характеризующуюся последовательностью SEQ ID NO: 2 или смысловую цепь, характеризующуюся последовательностью SEQ ID NO: 5 и антисмысловую цепь, характеризующуюся последовательностью SEQ ID NO: 6) и 10мМ цитратного буфера (буферизированного водного раствора) при рН 3,0;
и второго раствора, представляющего собой этанольную фазу, содержащую 4-[4-({[4-хлоро-3-(трифторометил)фенил]карбамоил}амино)фенокси]-N-метил-пиридин-2-карбоксамид или его фармацевтически приемлемую соль и липидную смесь (состоящую из ионизируемого липидоида C12-200, дистеароилфосфатидилхолина, холестерина, полиэтиленгликоля PEG2000_С14 в молярном соотношении (%) 45-50 : 10-20 : 35-38,5 : 0,4-1,5), растворенные в этаноле;
смешивание водной и этанольной фаз в соотношении 3:1 по объему со скоростью смешивания 10 мл/мин с получением липидных наночастиц, имеющих массовое отношение миРНК к ионизируемому липидоиду С12-200 - 1:5;
удаление этанола из смеси посредством диафильтрации относительно фосфатно-солевого буфера при pH 7,4;
стерилизация фильтрованием.
В некоторых вариантах изобретения стерилизацию осуществляют посредством фильтрации через PES фильтр с размером пор 0,22 мкм. Полученный водный раствор липидных наночастиц предпочтительно стерилизуют перед хранением или применением.
В предпочтительных вариантах изобретения молярное соотношение (%) компонентов липидной смеси: липидоида C12-200, дистеароилфосфатидилхолина, холестерина и полиэтиленгликоля PEG2000_С14 составляет 50:10:38.5:1,5.
Решение поставленной задачи также достигается путем применения предлагаемой фармацевтической композиции для лечения опухолевых заболеваний печени.
Решение поставленной задачи также достигается путем применения предлагаемой фармацевтической композиции для производства лекарственного средства для лечения опухолевых заболеваний печени.
Поставленная задача также решается путем разработки способа лечения опухолевых заболеваний печени у пациента, включающего введение пациенту предлагаемой фармацевтической композиции в терапевтически эффективных дозах и режимах.
В некоторых вариантах воплощения изобретения опухолевое заболевание представляет собой гепатоцеллюлярную карциному или гепатобластому.
В результате осуществления изобретения достигаются следующие технические результаты:
- разработаны гепатотропные липидные наночастицы, содержащие одновременно миРНК к ганкирину и сорафениб, и обеспечивающие повышение эффективности сорафениба в несколько (порядка девяти (!)) раз, при этом разработанные гепатотропные липидные наночастицы обеспечивают высокоэффективное инкапсулирование активных компонентов в липидные наночастицы и имеют оптимальный размер, достаточные для их эффективной доставки и воздействия на мишени действия;
- разработана принципиально новая лекарственная (фармацевтическая) композиция для лечения опухолевых заболеваний печени у пациента (таких как, например, гепатоцеллюлярная карцинома (ГЦК) и гепатобластома), наноструктура которой представляет собой гепатотропные липидные наночастицы, содержащие одновременно миРНК к ганкирину и сорафениб, и представляющая собой комбинацию классического подхода химиотерапии и новой клинически апробированной технологии РНК-интерференции;
- структура миРНК, входящих в композицию, обеспечивает эффективное достижение ими мишени действия за счет увеличенной стабильности миРНК к действию внутриклеточных нуклеаз, а также надежное блокирование синтеза ганкирина за счет структуры, обеспечивающей эффективную индукцию РНК-интерференции мРНК ганкирина; это позволяет использовать миРНК в низких концентрациях, что дополнительно уменьшает вероятность побочных эффектов для пациентов;
- предложенная фармацевтическая композиция обеспечивает ряд преимуществ по сравнению с другими вариантами комбинированного лечения, использующих сочетание известных противоопухолевых препаратов или же сочетание противоопухолевых препаратов с паллиативной терапией. Во-первых, воздействие на опухолевые клетки происходит на разных молекулярных уровнях: сорафениб взаимодействует с белками клеточного цикла опухолевых клеток, вызывая его остановку, а также регулирует множество сигнальных путей в опухолевой клетке, стимулируя запуск апоптоза, ингибирование же ганкирина на уровне РНК-интерференции приводит к практически полному отсутствию этого белка в опухолевой клетке, что дополнительно останавливает клеточное деление и усиливает действие сорафениба. Также, в связи с гетерогенной природой опухолей печени, предлагаемая лекарственная композиция позволяет обеспечить как комплексное, так и независимое терапевтическое действие входящих в нее активных компонентов, что позволяет преодолеть проблемы возникающей резистентности к сорафенибу, снизить терапевтическую дозу препарата химиотерапии и повысить эффективность терапии. Также стоит отметить, что ганкирин является селективным белком для опухолевых клеток печени, и практически отсутствует в нормальных гепатоцитах, таким образом, предлагаемая комбинаторная терапия уменьшает побочные эффекты сорафениба за счет снижения его эффективной дозы и увеличивает специфичность воздействия именно на раковые клетки печени.
Краткое описание рисунков
Фиг. 1. Проверка эффективности миРНК в экспериментах in vitro. (А) - Оценка уровня мРНК в культуре клеток Huh7 после трансфекции 10 нМ миРНК, специфичными к ганкирину. Уровень мРНК анализировали через 24 часа после трансфекции. (Б) и (В) - Кривые зависимости уровня мРНК ганкирина в культуре клеток Huh7 от концентрации миРНК №1 и №3, соответственно. Уровень мРНК анализировали через 24 часа после трансфекции (среднее значение ± SD). Р-критерий определяли методом однофакторного дисперсионного анализа, по сравнению с контролем (*P <0,05, ** P< 0,005, *** P< 0,0005). (Г) Оценка количества белка ганкирина в клетках Huh7 через 2, 4 и 7 дней после трансфекции миРНК №1 методом Вестерн-блот. Оценка количества тубулина использовалась в качестве референсного для оценки количества наносимого в гель материала.
Фиг. 2 Кривые оценки среднего размера ЛНЧ, содержащих миРНК с или без сорафениба. Обозначения: ЛНЧ1 - частицы, содержащие миРНК №1, ЛНЧ3 - частицы, содержащие миРНК №3; ЛНЧ(1+сораф) - частицы, содержащие миРНК №1, инкапсулированную совместно с сорафенибом; ЛНЧ(3+сораф) - частицы, содержащие миРНК №3, инкапсулированную совместно с сорафенибом.
Фиг. 3. Определение концентрации полумаксимального ингибирования сорафениба (IC50) с помощью МТТ-теста на линиях клеток карциномы (Huh7) и гепатомы (HepG2) человека. (А) - Определение IC50 при терапии только сорафенибом. (Б) - Определение IC50 в клетках Huh7 при совместной терапии сорафенибом и липидными наночастицами (ЛНЧ), содержащими миРНК. (В) - Определение IC50 в клетках HepG2 при совместной терапии сорафенибом и липидными наночастицами (ЛНЧ), содержащими миРНК; (Г) - Определение IC50 в клетках Huh7 при терапии сорафенибом, инкапсулированным в ЛНЧ совместно с миРНК. Обозначения на Фиг.3 соответствуют обозначениям на Фиг. 2.
Термины и определения
Если иное не оговаривается, все технические и научные термины, используемые в данной заявке, имеют то же самое значение, которое понятно для специалистов в данной области. Ссылки на методики, используемые при описании данного изобретения, относятся к хорошо известным методам, включая изменения этих методов и замену их эквивалентными методами, известными специалистам.
В документах данного изобретения термины «включает», «включающий» и т.п., а также «содержит», «содержащий» и т.п. интерпретируются как означающие «включает, помимо всего прочего» (или «содержит, помимо всего прочего»). Указанные термины не предназначены для того, чтобы их истолковывали как «состоит только из».
Термин «и/или» означает один, несколько или все перечисленные элементы.
Также здесь перечисление числовых диапазонов по конечным точкам включает все числа, входящие в этот диапазон.
Термины «лечение», «терапия» охватывают лечение патологических состояний у субъекта, предпочтительно у человека, и включают: а) блокирование (приостановку) течения заболевания, б) облегчение тяжести заболевания, т.е. индукцию регрессии заболевания.
Термины «субъект» и «пациент» охватывает все виды млекопитающих, предпочтительно человека.
Термин «предотвращение», «профилактика» охватывает случаи предотвращения рецидива состояния млекопитающего, характеризующегося злокачественной опухолью или метастазами злокачественной опухоли. К профилактической терапии относится, а) первичная профилактика и б) вторичная профилактика. Первичная профилактика определяется как профилактическое лечение у пациентов (субъектов), клиническая стадия заболевания у которых ещё не наступила. Вторичная профилактика - это предотвращение повторного наступления (рецидива) того же или близкого клинического состояния заболевания.
Под «эффективным количеством» («терапевтически эффективным количеством») активного вещества в фармацевтической композиции понимается количественное содержание активного вещества в композиции, достаточное для достижения терапевтического (и/или профилактического) эффекта у пациента. Поскольку фармацевтические композиции по изобретению содержат два терапевтически активных ингредиента, эффективное количество активных компонентов в составе композиции может быть рассчитано как по количеству сорафениба, необходимого для достижения терапевтического (или профилактического) эффекта, так и по количеству миРНК, необходимого для достижения эффекта. В одном из вариантов, но не ограничиваясь им, расчет может быть произведен, например, на основании известной рекомендуемой дозы сорафениба, с учетом его неожиданно повышенной (порядка 9 раз) эффективности воздействия на опухолевые клетки в составе композиции по изобретению. Данные об эффективном количестве активных ингредиентов в составе композиции также могут быть получены на основании результатов экспериментов, подробно представленных ниже, а также на основании дополнительных экспериментов.
Под «терапевтически эффективной дозой» подразумевается количество лекарственного средства, вводимого пациенту, при котором у него с наибольшей вероятностью проявится ожидаемый терапевтический эффект. Точное требуемое количество может меняться от субъекта к субъекту в зависимости от многочисленных факторов, таких как тяжесть заболевания, возраст, масса тела, общее состояние организма, комбинированное лечение с другими препаратами и др. Ведение лекарственного средства (фармацевтической композиции) по изобретению субъекту, нуждающемуся в лечении и/или профилактике заболевания или состояния, осуществляется в дозе, достаточной для достижения терапевтического эффекта. При проведении лечения и/или профилактики введение может осуществляться как разово, так и несколько раз, например несколько раз в неделю, несколько раз в месяц, чаще в виде курсового введения на протяжении времени, достаточного для достижения терапевтического эффекта (от нескольких дней до недели, нескольких недель и до месяцев), при этом курсы введения лекарственного средства могут проводиться повторно. В частности, при среднетяжелых формах заболевания, разовая доза, кратность и/или длительность введения препарата по изобретению могут быть увеличены. Схема лечения, а также длительность лечения также могут варьировать от пациента к пациенту и составлять от минимального курса лечения до проведения лечения курсами на протяжении всей жизни пациента. В процессе лечения схема приема препарата может корректироваться на основании наблюдаемых результатов, с использованием стандартных критериев эффективности, известных в данной области.
Фармацевтическая композиция (лекарственный препарат) по изобретению может быть введена в организм пациента парентеральным путем (в том числе внутривенно, путем капельной инфузии) в количестве, эффективным для лечения или профилактики заболевания.
Композиции по изобретению также могут вводиться в комбинации с другими известными агентами, применяемыми у пациентов с опухолями печени.
Термин «липидные наночастицы» («частицы», «ЛНЧ»)) относится к наночастицам, имеющим следующий состав: ионизируемый липидоид С12-200 (1,1'-((2-(4-(2-((2-(бис(2-гидроксидодецил)-амино)этил)(2-гидроксидодецил)амино)этил)пиперазин-1-ил)этил) азандиил) бис(додекан-2-ол)), дистеароилфосфатидилхолин (DSPC) (1,2-дистеароил-sn-глицеро-3-фосфохолин), холестерин, полиэтиленгликоль PEG2000-C14 (аммонийная соль 1,2-дистеароил-sn-глицеро-3-фосфоэтаноламин-N-[амино(полиэтиленгликоля)-2000]) в молярном соотношении (%) 45-50 : 10-20 : 35-38,5 : 0,4-1,5, в более предпочтительных вариантах - в соотношении 50 : 10 : 38.5 : 1,5. ЛНЧ на основе липидоида С12-200 хорошо известны в уровне техники для доставки миРНК (см., например, Love K.T et al. Lipid-like materials for low-dose, in vivo gene silencing // Proc Natl Acad Sci U S A . 2010 Feb 2;107(5):1864-9). Как показали проведенные эксперименты, липидные наночастицы по изобретению имеют средний диаметр приблизительно 60-130 нм, более часто приблизительно 70-110 нм, наиболее часто приблизительно 70-90 нм.
Фармацевтические композиции по изобретению представляют собой стерильную композицию миРНК и сорафениба, совместно инкапсулированных в ЛНЧ, в водной фазе (воде). Количество воды выбирается достаточным для формирования липидных наночастиц по изобретению.
«Вода» («водная фаза») в рамках настоящего изобретения представляет собой «подготовленную воду» - деионизированную, дистиллированную, очищенную, кондиционированную воду, в том числе воду для инъекций, получаемую технологической водоподготовкой в ходе изготовления лекарственных средств, медицинских изделий, косметики.
«Инкапсулированный» («полностью инкапсулированный») при использовании в контексте изобретения означает, что молекулы миРНК в липидных наночастицах не подвергаются существенной деградации после воздействия сыворотки крови или в ходе анализа с применением нуклеаз, что вызвало бы существенную деградацию свободной РНК. В полностью инкапсулированной системе предпочтительно менее 25% миРНК в частице деградирует в условиях обработки, при которых в норме было бы деградировано 100% свободной нуклеиновой кислоты, более предпочтительно - менее 10% и наиболее предпочтительно - менее 5% нуклеиновой кислоты в частице деградирует. Полное инкапсулирование также предполагает, что частицы устойчивы к действию сыворотки, т.е. что они не претерпевают быстрое разрушение на компоненты после введения in vivo.
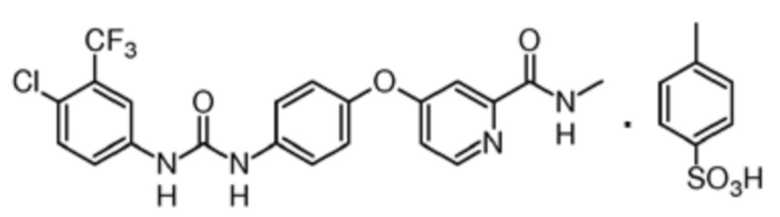
Сорафениб (4-[4-[[4-хлор-3-(трифторметил)фенил]карбамоиламино]фенокси]-N-метилпиридин-2-карбоксамид) - мультикиназный ингибитор. Показано, что сорафениб подавляет, по меньшей мере, многочисленные внутриклеточные киназы с-CRAF, BRAF и мутантную BRAF, и киназы, расположенные на поверхности клетки KIT, FLT-3, RET, VEGFR-1, VEGFR-2, VEGFR-3 и PDGFR-β, по крайней мере частично задействованные в сигнальных системах опухолевой клетки, в процессах ангиогенеза и апоптоза. Для целей настоящего изобретения используют сорафениб в форме свободного основания или в виде фармацевтически приемлемой соли. Используемый здесь термин «фармацевтически приемлемый» относится к таким соединениям, которые, в рамках проведенного медицинского заключения, пригодны для использования в контакте с тканями человека без излишней токсичности, раздражения, аллергической реакции и т.д., и отвечают разумному соотношению пользы и риска. При этом «фармацевтически приемлемая соль» относится к обычным нетоксичным солям, образующимся путем реакции молекулы сорафениба с неорганическими или органическими кислотами. При этом неорганические кислоты включают, например, хлористоводородную кислоту, бромистоводородную кислоту, аминосульфоновую кислоту, и т.д., органические кислоты включают винную кислоту, молочную кислоту, бензолсульфоновую кислоту, п-толуолсульфоновую кислоту, метансульфоновую кислоту, нафталинсульфоновую кислоту, этансульфоновую кислоту, нафталиндисульфоновую кислоту, малеиновую кислоту, гидроксималеиновую кислоту, и т.д. В частных вариантах изобретения фармацевтически приемлемая соль сорафениба представляет собой соль с п-толуолсульфоновой кислотой (тозилат или геми-тозилат моногидрат). В предпочтительных вариантах изобретения фармацевтически приемлемая соль сорафениба представляет собой сорафениб тозилат (4-[4-[[4-хлор-3-(трифторметил)фенил]карбамоиламино]фенокси]-N-метилпиридин-2-карбоксамид, 4-метилбензолсульфоновая кислота), имеющий следующую структурную формулу:
 .
.
Если не определено отдельно, технические и научные термины в данной заявке имеют стандартные значения, общепринятые в научной и технической литературе.
Подробное описание изобретения
Настоящее изобретение направлено на разработку нового лекарственного препарата, эффективного для лечения опухолевых заболеваний печени, а также на разработку метода лечения указанных заболеваний с использованием такого препарата.
Высокая эффективность разработанного препарата основана на комбинации двух подходов к лечению опухолевых заболеваний печени: классической химиотерапии сорафенибом и принципиально нового типа терапии с использованием РНК-интерференции, обеспечивающим подавление экспрессии ганкирина, регулирующего пролиферацию опухолевых клеток.
Как уже отмечалось, экспрессия ганкирина практически отсутствует в нормальных гепатоцитах, слабо выражена при циррозе и порокарциноме печени и наиболее явно представлена в печени при опухолевых заболеваниях печени, в том числе ГЦК. При этом известно, что ганкирин может способствовать прогрессированию и метастазированию опухолей печени. Поэтому снижение уровня ганкирина в опухолевых клетках является перспективным методом лечения опухолевых заболеваний печени.
Одним из новых эффективных методов снижения продукции целевого белка, синтезируемого клеткой, является метод РНК-интерференции, основанный на селективном подавлении экспрессии генов за счет деградации мРНК, опосредованной малыми интерферирующими РНК (миРНК).
Нижеследующие примеры приведены в целях раскрытия характеристик настоящего изобретения, и их не следует рассматривать как каким-либо образом ограничивающие объем изобретения.
Линии клеток и трансфекция
Линии клеток Huh7 (карцинома) и HepG2 (гепатома) человека (ATCC, Manassas, VA) выращивали в среде DMEM/F12, с добавлением 10% фетальной бычьей сывороткой (FBS) (Gibco, Waltham, MA), 2 мМ L-глютамина (Gibco) и 1% пенициллин-стрептомицина при 37°С и 5% СО2. Работа с клетками проводилась при конфлюентности 80%. миРНК (10 нМ или 1 нМ) к ганкирину и миРНК против люциферазы (контроль) трансфицировали с использованием Lipofectamine RNAimax (Invitrogen, Waltham, MA) в соответствии с инструкциями производителя.
Статистический анализ
Prism 7 (GraphPad Software, La Jolla, CA) использовали для статистического анализа. Двусторонние непарные тесты Манна-Уитни с 95% доверительными границами использовались для статистического анализа. Значение р<0,05 считали значимым.
Пример 1. Разработка и селекция миРНК
В рамках настоящего изобретения для подавления экспрессии мРНК ганкирина человека был проведен дизайн миРНК на основании последовательности мРНК человеческого ганкирина (номер NCBI GenBank: NM_002814.4) и известных критериев по отбору, с исключением возможных участков связывания эндогенных микроРНК, а также последовательностей, встречающихся в других РНК транскриптома человека. Последовательности 19-звенных кандидатов были сопоставлены с базой данных мРНК (RefSeq) и оценены по их способности связываться вне целевой мишени. В частности, миРНК были ранжированы на основе количества/положений несоответствий в разных частях и несоответствий в положении сайта расщепления. Полученные последовательности дополнительно проверяли, чтобы исключить наличие известных мотивов микроРНК и мотивов иммуностимулирующих последовательностей.
Для увеличения стабильности миРНК к действию внутриклеточных нуклеаз в процессе синтеза были использованы 2'-О-метильные пиримидиновые нуклеотиды и введены тиофосфатные группы между двумя последними нуклеотидами с 3'-конца. Введение 2'-O-метил (2'-OMe) пиримидиновых нуклеотидов в миРНК дополнительно снижает иммунный ответ и возможность неспецифичного действия, а вместе с использованием 3'-межнуклеотидных тиофосфатов повышает стабильность против нуклеаз.
В результате проведенных исследований был разработан дизайн шести миРНК, потенциально активных в отношении подавления экспрессии мРНК ганкирина (см. Таблицу 1).
Примечание: заглавные буквы прямого начертания - рибонуклеотиды; заглавные курсивные буквы - дезоксирибонуклеотиды; строчные буквы - 2'-O-метилрибонуклеотиды; s - тиофосфатная связь.
Контрольная миРНК - миРНК к гену люциферазы светлячка.
Ниже представлено соответствие нуклеотидных последовательностей, представленных в Таблице 1, их номерам в Перечне последовательностей согласно стандарту ST.26, являющемуся частью настоящего описания изобретения. В случае наличия разночтений в структуре последовательностей между Таблицей 1 и соответствующей последовательностью в Перечне последовательностей согласно стандарту ST.26, приоритетными являются данные, представленные в Таблице 1.
как она представлена в Таблице 1
миРНК были синтезированы по амидофосфитной схеме (MerMade 12, BioAutomation, США), очищены методом ионообменной хроматографии, а их чистоту подтверждали методом ВЭЖХ-МС. В качестве контрольной миРНК использовали миРНК к гену люциферазы светлячка. Активность синтезированных миРНК была проверена в клеточной линии карциномы печени Huh7 методом ПЦР с обратной транскрипцией (ОТ-ПЦР) (см. Фиг. 1А).
Для выделения общей РНК клетки лизировали с использованием реагента TRIzol (Invitrogen) с последующим осаждением изопропанолом в соответствии с инструкциями производителя. кДНК получали с использованием набора для обратной транскрипции кДНК Maxima First Strand kit (Thermo Scientific). Уровень мРНК ганкирина оценивали с помощью наборов для проведения ПЦР-РВ с использованием SYBR Green (Thermo Fisher Scientific) в термоциклере CFX96 (Bio-rad). Количества мРНК были нормализованы с использованием мРНК глицеральдегид-3-фосфатдегидрогеназы (GAPDH) человека. Список праймеров приведен в Таблице 3.
Несмотря на то, что дизайн синтезированных миРНК был тщательно проработан и теоретически все шесть миРНК (№1 - №6) должны эффективно подавлять синтез ганкирина, проведенные эксперименты показали, что только миРНК №1 и №3 способны практически полностью подавлять экспрессию мРНК, а следовательно, и блокировать синтез ганкирина. Как видно на Фиг.1А, остаточное количество мРНК ганкирина в случае использования миРНК №1 и №3, в отличие от других миРНК, составило менее 15%.
Таким образом, в результате проведённых исследований были разработаны две наиболее активные миРНК (№1 и №3), для которых в дальнейшем были определены концентрации полумаксимального ингибирования (IC50). Для этого клетки Huh7 обрабатывали различными концентрациями миРНК №1 и №3 в диапазоне от 0,00017 до 20 нМ, после чего выделяли суммарную РНК и анализировали эффективность снижения уровня мРНК ганкирина методом ПЦР-РВ (см. Фиг.1Б и 1В). Как показали проведенные эксперименты, значения концентрации полумаксимального ингибирования (IC50) составляют 1,89±0,12 пМ (для миРНК №1) и 9,49±0,02 пМ (для миРНК №3). Такие пикомолярные концентрации IC50 свидетельствуют о неожиданно высокой эффективности полученных миРНК. Это позволяет использовать разработанные миРНК в низких концентрациях, что дополнительно уменьшает вероятность побочных эффектов для пациентов.
Дополнительно была исследована динамика снижения концентрации белка ганкирина в клетках Huh7 методом РНК-интерференции во времени. Для этого клетки трансфецировали 1 нМ концентрацией миРНК №1 и анализировали количество белка ганкирина на 2-й, 4-й и 7-й день после трансфекции (см. Фиг. 1Г). Клетки лизировали в буфере RIPA, содержащем полный набор ингибиторов протеаз (Roche). Экстракты инкубировали 30 минут на льду и центрифугировали при 12000 g в течение 20 минут при 4°С. Концентрации общего белка в супернатантах определяли с использованием бицинхониновой кислоты (набор реагентов BCA, Pierce) по методике производителя. Далее образцы анализировали методом денатурирующего гель-электрофореза по Лэммли (10%), с общим количеством белка 20 мг в дорожку. Разделенные белки анализировали вестерн-блоттингом с использованием следующих антител: анти-tubulin (Cell Signaling) (для анализа контроля), анти-Gankyrin (Abcam) (для анализа ганкирина). Вестерн-блоты визуализировали с использованием реагента Clarity Max Western ECL Substrate (Bio-rad) в соответствии с инструкциями производителя. Проведенные исследования показали, что уже на второй день после добавления миРНК количество белка ганкирина плохо детектируется, а эффективность ингибирования составляет более 80%.
Пример 2. Получение липидных наночастиц, содержащих только миРНК или миРНК в комбинации с сорафенибом
Частицы готовили на микрофлюидном чипе смешением водной и этанольной фаз. В качестве водной фазы использовали раствор миРНК (№1 или №3 из примера 1) (0,1 мг/мл) в 10мМ цитратном буферном растворе (рН 3.0); в качестве этанольной - раствор липидов, соответственно, с добавлением или без добавления сорафениба тозилата. Липидная смесь содержала следующие компоненты: ионизируемый липидоид C12-200, дистеароилфосфатидилхолин (DSPC) (Avanti Polar Lipids, США), холестерин (SigmaAldrich, США), полиэтиленгликоль PEG2000_С14 (Avanti Polar Lipids, США) в молярном соотношении (%) 45-50:10-20:35-38,5:0,4-1,5. Для добавления сорафениба тозилата предварительно получали его спиртовой раствор (5 мг/мл); в этанольную фазу добавляли 40,3 мкл полученного раствора.
Для формирования частиц требуемого размера водную и этанольную фазы смешивали в соотношении 3 : 1 по объему с общей скоростью смешивания 10 мл/мин с использованием микрофлюидного устройства NanoAssemblr™ Benchtop (Precision Nanosystems, США). Количество липидов на единицу миРНК рассчитывали из массового соотношения миРНК к ионизируемому липидоиду С12-200 - 1:5.
Полученную водно-спиртовую суспензию частиц разбавляли стандартным фосфатно-солевым буфером, далее проводили диализ в 500 объемах стандартного фосфатно-солевого буфера при pH 7,4 в диализных кассетах с мембраной, отделяющей соединения с молекулярной массой 20000 Да (MW) (66012; Thermo Fisher Scientific) в течение 18 часов, для удаления этанола и замены буфера. После диализа частицы фильтровали в стерильных условиях через шприцевой фильтр с мембраной из полиэфирсульфона (PES) (размер пор 0,22 мкм) (3915; Corning) и хранили при +4°C.
После диализа определяли загрузку частиц по миРНК и сорафенибу, а также измеряли размеры полученных частиц.
Определение загрузки миРНК. Концентрацию загруженной в липидные наночастицы миРНК определяли по разнице значений уровня флуоресцентного сигнала при окрашивании реагентом RiboGreen (R11491; Thermo Fischer Scientific, США) суспензии частиц до их разрушения и после. Для разрушения частиц использовали детергент Triton X-100 (Sigma-Aldrich, США).
Определение загрузки сорафениба. Содержание сорафениба в частицах определяли с помощью калибровочной кривой по поглощению на 266 нм в этаноле. Так как присутствие миРНК мешает определению загрузки сорафениба на используемой длине волны, при определении загрузки предварительно осаждали содержавшуюся в частицах миРНК. Для этого к аликвоте частиц добавляли 96% спирт и оставляли при -80°С на ночь. Затем осадок миРНК центрифугировали на 13 000 об/мин при +4°С, супернатант осторожно отбирали и проводили измерение оптической плотности на 266 нм.
Измерения размера частиц проводили с использованием Zetasizer Nano ZSP (Malvern Panalytical) в соответствии с протоколом производителя (Таблица 4). Измерения зета-потенциала проводили в условиях нейтрального рН. Указанные значения являются средней величиной для 10-25 повторов.
в ЛНЧ, мкМ
Данные о размерах полученных ЛНЧ, содержащих липидоид С12-200, дистеароилфосфатидилхолин, холестерин и PEG2000-C14 в молярном соотношении (%) 50 : 10 : 38.5 : 1,5, приведены на Фиг.2. Контроль представляет собой липидные наночастицы, содержащие миРНК к мРНК люциферазы светлячка, используемые для сравнения размера и загрузки частиц. Все полученные частицы имеют распределение размеров в пределах 100 нм, что является оптимальным для применения в качестве контейнеров для доставки лекарственных препаратов, так как частицы большего размера активируют лимфатическую систему и быстрее выводятся из кровообращения (см., например, книгу Drug delivery nanoparticles formulation and characterization под редакцией Y. Pathak, D. Thassu, 2009). Как показали проведенные измерения, эффективность загрузки миРНК в ЛНЧ составляет 65-85%, что свидетельствует о высокоэффективном инкапсулировании. При совместном включении в ЛНЧ сорафениба и миРНК эффективность загрузки миРНК в ЛНЧ составила порядка 55%, при этом содержание миРНК в липидных наночастицах по отношению к сорафенибу составило 5-8 мол.%. Полученные данные свидетельствуют о высокоэффективном инкапсулировании активных компонентов, достаточным для их эффективной доставки и воздействия на мишени.
Пример 3. Исследование активности комбинации миРНК с сорафенибом на клеточных линиях
В качестве характеристики активности для сорафениба была исследована концентрация полумаксимального ингибирования в двух клеточных линиях человека - Huh7 (карцинома) и HepG2 (гепатома) МТТ-тестом. Для этого к клеткам добавляли возрастающую концентрацию сорафениба тозилата от 0 до 20 мкM и анализировали жизнеспособность клеток через 48 часов. Концентрации полумаксимального ингибирования (IC50) для сорафениба тозилата в клеточных линиях карциномы и гепатомы человека оказались близкими по значению и равны 2,23±0,34 и 2,41±0,38 мкМ (см. Фиг. 3А).
В следующем эксперименте клетки гепатомы (HepG2) и карциномы (Huh7) человека трансфицировали 1 нМ ЛНЧ, включающих миРНК к ганкирину, согласно изобретению, и инкубировали два дня (было показано, что за это время происходит существенное снижение количества ганкирина в клетках). Для определения IC50 сорафениба при проведении комбинированного воздействия, к трансфицированным клеткам добавляли возрастающую концентрацию сорафениба тозилата от 0 до 20 мкM и анализировали жизнеспособность клеток через 48 часов МТТ-тестом.
Проведенные измерения показали, что комбинированное использование сорафениба совместно с ЛНЧ, включающими миРНК, приводит к снижению концентрации полумаксимального ингибирования в обеих клеточных линиях в два раза (и составило 1,24±0,12 мкМ для карциномы и 1,16±0,13 мкМ для гепатомы в случае миРНК №1; 1,54±0,21 мкМ для карциномы и 1,64±0,31 мкМ для гепатомы в случае миРНК №3 - см.Фиг.3Б и Фиг.3В). Таким образом, как видно из результатов проведенного исследования, в случае комбинированной терапии пролиферация опухолевых клеток ингибируется при более низкой концентрации сорафениба, что свидетельствует о том, что применение сорафениба в комбинации с миРНК к ганкирину по изобретению существенно повышает эффективность его воздействия на опухолевые клетки. Такая комбинация позволяет использовать сорафениб в существенно более низких дозах, что, в свою очередь, снижает риск побочных эффектов для пациентов.
Однако еще более неожиданные результаты были получены в следующем эксперименте, когда была исследована эффективность комбинированной терапии сорафенибом и миРНК к ганкирину, совместно инкапсулированными в липидные наночастицы. Для этого клетки карциномы человека (Huh7) трансфицировали от 0 до 5 мкM ЛНЧ по изобретению и анализировали жизнеспособность клеток через 3 дня методом МТТ-теста. Проведенные измерения показали (см. Фиг.3Г), что комбинированное использование сорафениба совместно с миРНК, инкапсулированными совместно в ЛНЧ, приводит к снижению концентрации полумаксимального ингибирования не в два, а в девять раз (0,262±0,015 мкМ для ЛНЧ(1+сораф) и 0,323±0,057 мкМ для ЛНЧ(3+сораф)) по сравнению с чистым сорафенибом (2,41±0,28 мкМ), из чего следует, что включение сорафениба в состав ЛНЧ повышает эффективность воздействия на опухолевые клетки печени почти в пять раз.
Таким образом, в результате проведенных исследований было установлено, что включение сорафениба в ЛНЧ совместно с миРНК к ганкирину №1 или №3 по изобретению приводит к гораздо более существенному повышению эффективности ингибирования пролиферации опухолевых клеток, чем его применение вне ЛНЧ с миРНК. Такое существенное повышение эффективности терапии позволяет использовать сорафениб в значительно меньших концентрациях, что снижает токсическую нагрузку химиотерапевтического агента и риск побочных эффектов для пациентов.
Таким образом, настоящее изобретение представляет собой комбинацию классического подхода химиотерапии и новой клинически апробированной технологии РНК-интерференции. Разработанная фармацевтическая композиция эффективно подавляет пролиферацию опухолевых клеток печени, способствуя лечению онкологических заболеваний. Использование нового подхода в несколько раз усиливает действие химиотерапевтического препарата сорафениба при лечении опухолевых заболеваний, что позволяет использовать сорафениб в значительно меньших концентрациях и тем самым существенно снизить токсическую нагрузку и риск развития побочных эффектов у пациентов.
Несмотря на то, что изобретение описано со ссылкой на раскрываемые варианты воплощения, для специалистов в данной области должно быть очевидно, что конкретные подробно описанные эксперименты приведены лишь в целях иллюстрирования настоящего изобретения, и их не следует рассматривать как каким-либо образом ограничивающие объем изобретения. Должно быть понятно, что возможно осуществление различных модификаций без отступления от сути настоящего изобретения.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="siRNA-18 (3).xml"
softwareName="WIPO Sequence" softwareVersion="2.0.0"
productionDate="2022-08-30">
<ApplicantFileReference>474750</ApplicantFileReference>
<ApplicantName languageCode="ru">АВТОНОМНАЯ НЕКОММЕРЧЕСКАЯ
ОБРАЗОВАТЕЛЬНАЯ ОРГАНИЗАЦИЯ ВЫСШЕГО ОБРАЗОВАНИЯ «СКОЛКОВСКИЙ ИНСТИТУТ
НАУКИ И ТЕХНОЛОГИЙ»</ApplicantName>
<ApplicantNameLatin>SKOLKOVO INSTITUTE OF SCIENCE AND TECHNOLOGY
(SKOLTECH))</ApplicantNameLatin>
<InventionTitle languageCode="ru">КОМБИНИРОВАННАЯ ТЕРАПИЯ
ГЕПАТОЦЕЛЛЮЛЯРНОЙ КАРЦИНОМЫ НА ОСНОВЕ СОРАФЕНИБА</InventionTitle>
<SequenceTotalQuantity>18</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>20^21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q2">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thiophosphate bond</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>2..3</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>13..14</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>20..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thymidine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gccgggatgagattgtaaatt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>20^21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q6">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thiophosphate bond</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>20..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q7">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thymidine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>3</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tttacaatctcatcccggctt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>20^21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q10">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thiophosphate bond</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>20..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q54">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thymidine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>1..3</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>10</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>14</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>16..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cccaaggagcaagtatttatt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q13">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>20^21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q14">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thiophosphate bond</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>20..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q15">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thymidine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>taaatacttgctccttgggtt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>20^21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q17">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thiophosphate bond</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>20..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thymidine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>10</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>14</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caatcaaaatggctgtacttt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q19">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>20^21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q20">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thiophosphate bond</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>20..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q21">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thymidine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>3</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agtacagccattttgattgtt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>20^21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q23">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thiophosphate bond</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>20..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thymidine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>6..7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>10</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>14</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagtgccagtgaatgataatt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q25">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>20^21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q26">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thiophosphate bond</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>20..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q27">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thymidine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttatcattcactggcactctt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>20^21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q29">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thiophosphate bond</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>20..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thymidine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>5..6</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>10</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>14</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctaacctaatggtctgcaatt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q31">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>20^21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q32">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thiophosphate bond</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>20..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q33">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thymidine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>12</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttgcagaccattaggttagtt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>20^21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q35">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thiophosphate bond</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>20..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q36">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thymidine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>14..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>17..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cagatgctaaggaccattatt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q37">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>20^21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q38">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thiophosphate bond</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>20..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q39">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thymidine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>14</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>taatggtccttagcatctgtt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q40">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>20^21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q41">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thiophosphate bond</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>20..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q42">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thymidine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q43">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>2'-O-methyladenosine</INSDQualifier_v
alue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>6</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>gm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>10</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q44">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>2'-O-methyladenosine</INSDQualifier_v
alue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>12</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>14</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>gm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cttacgctgagtacttcgatt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q45">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>20^21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q46">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thiophosphate bond</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>20..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q47">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>thymidine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>3</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>gm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q48">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>2'-O-methyladenosine</INSDQualifier_v
alue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>um</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>cm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>gm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>gm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q49">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>2'-O-methyladenosine</INSDQualifier_v
alue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>gm</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgaagtactcagcgtaagtt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q50">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgcaccaccaactgcttagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q51">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggcatggactgtggtcatgag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q52">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cactggcactccaagttgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q53">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctggccgataaatccctgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Набор реагентов для определения малых рибонуклеиновых кислот (микроРНК) при диагностике множественной миеломы | 2024 |
|
RU2836054C1 |
| Способ получения мышиной модели для изучения миодистрофии Дюшенна и вариантов ее терапии | 2023 |
|
RU2815936C1 |
| Способ формирования системы иммунного надзора за опухолевыми клетками в организме млекопитающих | 2024 |
|
RU2828654C1 |
| Способ модификации антисмыслового олигонуклеотида для онкомаркер-зависимой деградации целевой мРНК | 2024 |
|
RU2839284C1 |
| Нуклеиновая кислота, содержащая регуляторные элементы H4C2, AES и mtRNR1 | 2023 |
|
RU2831168C1 |
| Нуклеиновая кислота, содержащая регуляторные элементы TPL, AES и mtRNR1 | 2023 |
|
RU2831163C1 |
| Нуклеиновая кислота, содержащая регуляторные элементы гена кроличьего β-глобина, mtRNR1 и AES | 2023 |
|
RU2831164C1 |
| Нуклеиновая кислота, содержащая регуляторные элементы гена кроличьего β-глобина, mtRNR1 и EMCV | 2023 |
|
RU2831165C1 |
| ПЕПТИД, ПРИМЕНЯЕМЫЙ ДЛЯ ИММУНОТЕРАПЕВТИЧЕСКИХ СРЕДСТВ | 2021 |
|
RU2836172C1 |
| Рекомбинантная плазмидная ДНК, кодирующая микроРНК hsa-miR-145-5p, и ее применение в качестве положительного контроля при ПЦР-РВ для диагностики сердечно-сосудистых заболеваний и выявления риска их развития | 2025 |
|
RU2837874C1 |
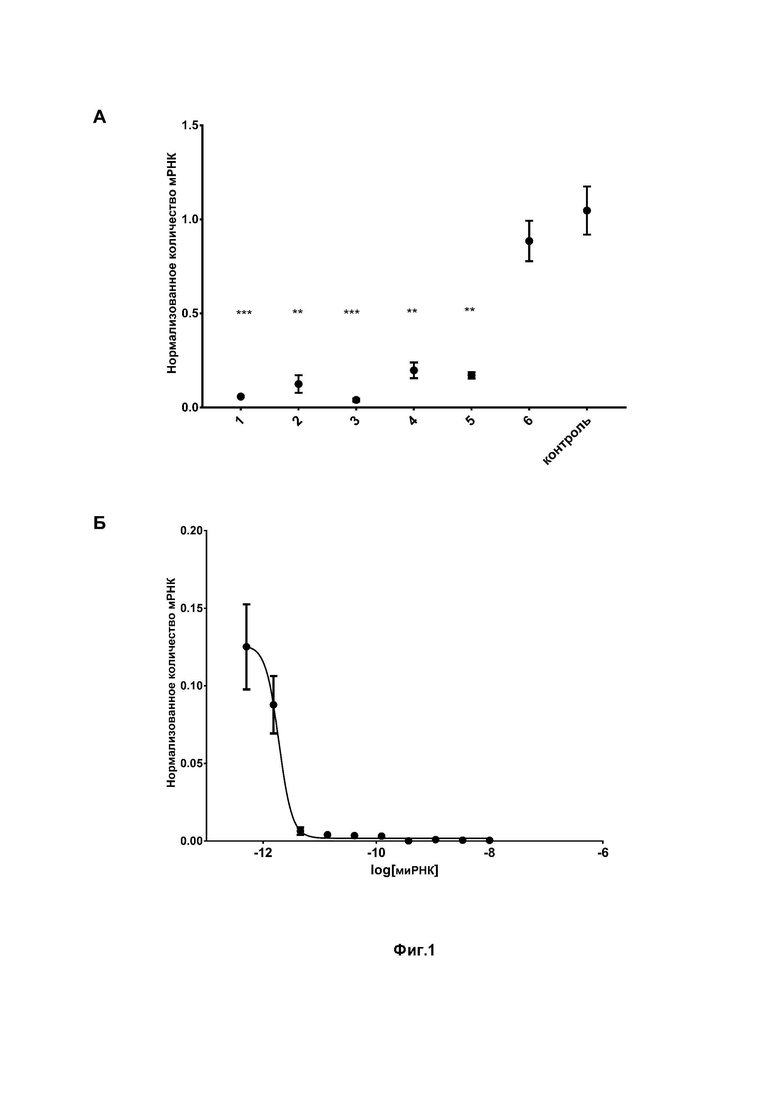

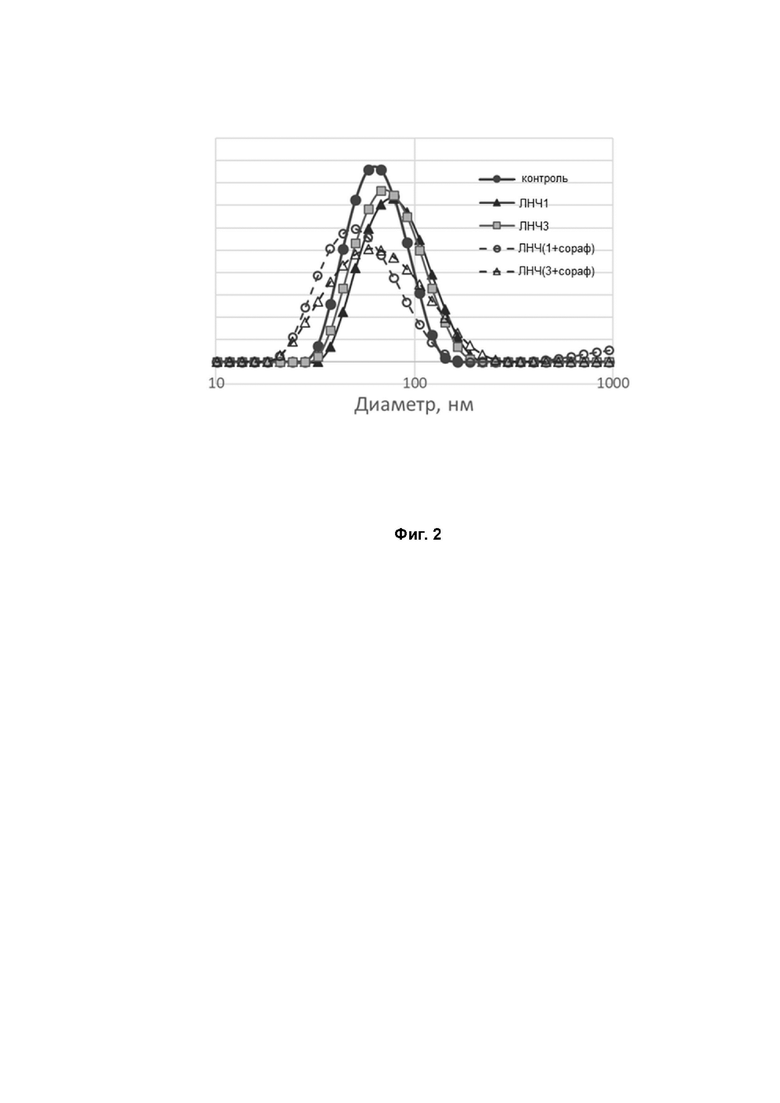
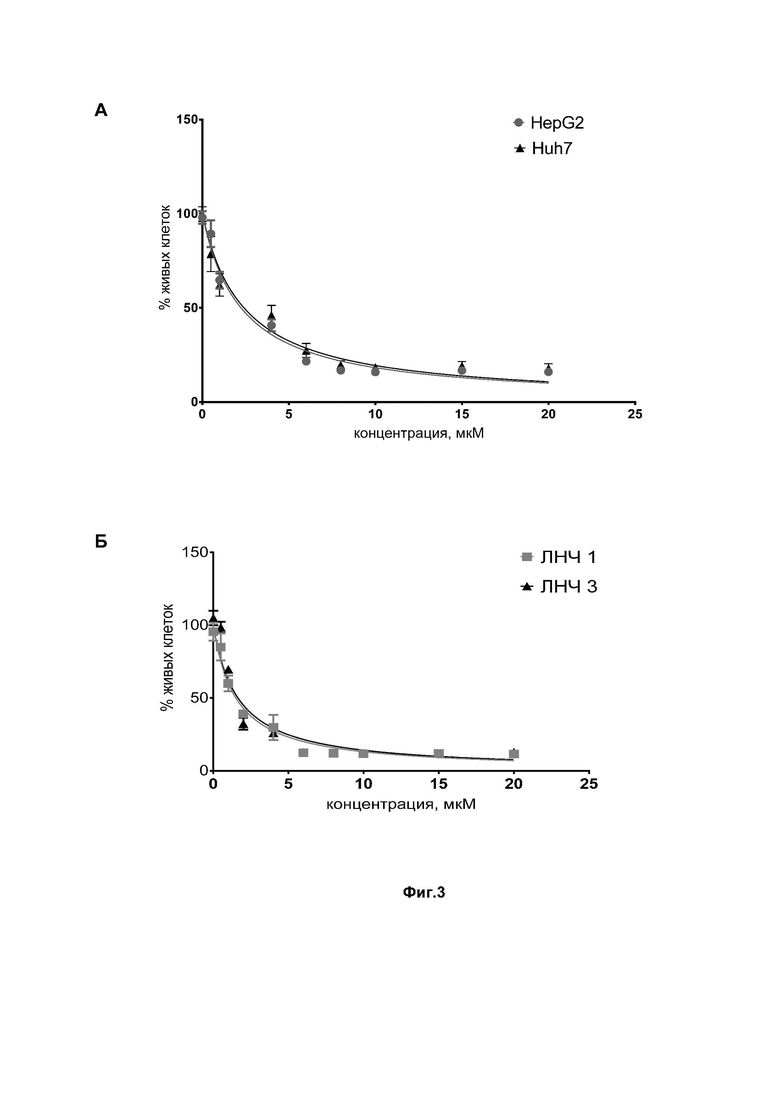
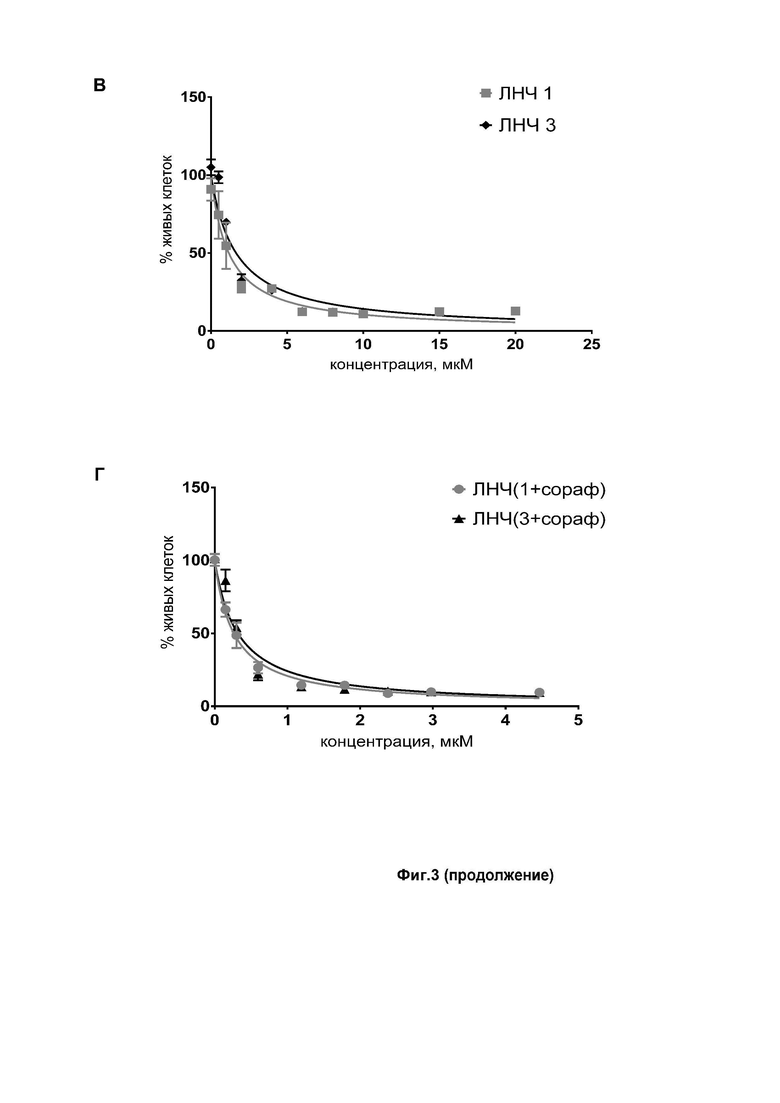
Изобретение относится к области биотехнологии. Описана группа изобретений, включающая липидную наночастицу для лечения опухолевых заболеваний печени у пациента, фармацевтическую композицию для лечения опухолевого заболевания печени у пациента и применение фармацевтической композиции для производства лекарственного средства для лечения опухолевого заболевания печени у пациента. Изобретение расширяет арсенал средств для лечения опухолевых заболеваний печени у пациента. 3 н. и 4 з.п. ф-лы, 3 ил., 4 табл., 3 пр.
1. Липидная наночастица для лечения опухолевых заболеваний печени у пациента, содержащая
(1) малую интерферирующую РНК (миРНК), способную ингибировать экспрессию ганкирина, где миРНК представляет собой дуплекс, содержащий
а) смысловую цепь, имеющую последовательность SEQ ID NO: 1, и антисмысловую цепь, имеющую последовательность SEQ ID NO: 2; или
б) смысловую цепь, имеющую последовательность SEQ ID NO: 5, и антисмысловую цепь, имеющую последовательность SEQ ID NO: 6,
и
(2) 4-[4-({[4-хлоро-3-(трифторометил)фенил]карбамоил}амино)фенокси]-N-метил-пиридин-2-карбоксамид или его фармацевтически приемлемую соль,
инкапсулированные совместно в вышеуказанные липидные наночастицы, при этом вышеуказанные липидные наночастицы содержат в молярном соотношении: 50% липидоида С12-200, 10% дистеароилфосфатидилхолина, 38,5% холестерина и 1,5% полиэтиленгликоля PEG2000-C14.
2. Липидная наночастица по п.1, в которой содержание компонента (1) по отношению к компоненту (2) составляет 5-8 мол.%.
3. Липидная наночастица по п.1, в которой фармацевтически приемлемая соль 4-[4-({[4-хлоро-3-(трифторометил)фенил]карбамоил}амино)фенокси]-N-метил-пиридин-2-карбоксамида представляет собой тозилат.
4. Фармацевтическая композиция для лечения опухолевого заболевания печени у пациента, включающая липидную наночастицу по любому из пп.1-3 в эффективном количестве и фармацевтически приемлемый носитель, представляющий собой воду.
5. Фармацевтическая композиция по п.4, в которой вода представляет собой деионизованную воду или воду для инъекций.
6. Применение фармацевтической композиции по любому из пп.4, 5 для производства лекарственного средства для лечения опухолевого заболевания печени у пациента.
7. Применение по п.7, в котором указанное опухолевое заболевание представляет собой гепатоцеллюлярную карциному или гепатобластому.
| Тест-классификатор клинического ответа на лечение сорафенибом индивидуальных пациентов с раком почки | 2018 |
|
RU2747746C2 |
| ФАРМАЦЕВТИЧЕСКИЙ ПРЕПАРАТ В ФОРМЕ ТАБЛЕТКИ И СПОСОБ ЕГО ПОЛУЧЕНИЯ | 2019 |
|
RU2747525C2 |
| WO 2011128403 A1, 20.10.2011 | |||
| WO 2010128259 A1, 11.11.2010. | |||
Авторы
Даты
2023-07-17—Публикация
2022-09-01—Подача