Область техники, к которой относится изобретение.
Изобретение относится к сельскому хозяйству и биотехнологии и может быть использовано для отбора подсолнечника, устойчивого к расе G заразихи с помощью проведения полимеразной цепной реакции.
Уровень техники.
Заразиха кумекая, или подсолнечная (Orobanche cumana Wallr.) - облигатный паразит подсолнечника, относящийся к высшим травянистым растениям. Широко распространен в большинстве стран мира, возделывающих эту культуру, и является одним из главных ограничивающих факторов в получении высоких урожаев [Антонова Т.С. Заразиха на подсолнечнике. - Краснодар: Просвещение-Юг, 2018. - 58 с.]. К настоящему времени обнаружено 8 рас этого патогена: А, В, С, D, Е, F, G и Н. Одной из наиболее вирулентных и распространенных из них на данный момент является раса G. Заразиха угнетает растения подсолнечника, отбирая у них питательные вещества и воду. У пораженных заразихой растений подсолнечника наблюдается потеря листьями тургора, увядание в результате дефицита воды, пожелтение и их частичное засыхание, а в засушливые годы - гибель растений. Для снижения негативных последствий, вызываемых паразитом, применяются различные дорогостоящие агротехнологические приемы, в особенности обработка посевов гербицидами. Возделывание же устойчивых сортов и гибридов подсолнечника позволит более экономно и экологически безопасно противодействовать заразихе [Лукомец В.М. Современные тренды селекционно-генетического улучшения сортов и гибридов подсолнечника во ВНИИМК / В.М. Лукомец, М.В. Трунова, Я.Н. Демурин // Вавиловский журнал генетики и селекции. - 2021. - Т. 25. - №4. -С.388-393].
Классический отбор генотипов подсолнечника по признаку устойчивости к заразихе проводится по результатам фенотипической оценки на инфекционном фоне [Антонова Т.С. Отбор на устойчивость к расе G заразихи из расщепляющихся популяций подсолнечника в искусственных условиях выращивания / Т.С. Антонова, Е.А. Стрельников, Н.М. Арасланова, С.З. Гучетль, Т.А. Челюстникова // Масличные культуры. Научно-технический бюллетень Всероссийского научно-исследовательского института масличных культур. -2017. - №3 (171). - С. 18-22]. Собранные с разных полей семена подсолнечника (каждый образец отдельно) используют для создания жестких инфекционных фонов в цветочных ящиках размером 20 × 17 × 90 см. Ящики заполняют почвенно-песчаной смесью в соотношении 1:1, смешанной с семенами заразихи из расчета 200 мг на 1 кг, и высевают семена линий дифференциаторов подсолнечника, известных своей устойчивостью к определенным расам. Растения выращивают в тепличных условиях при температуре 25-27°С и 16-часовом фотопериоде с дополнительным освещением в утренние и вечерние часы, а также в пасмурные дни. Выкапывают растения подсолнечника через 30-35 дней после появления всходов и с помощью подсчета количества клубеньков и побегов заразихи на корнях определяют расы заразихи, присутствующие в популяции [Антонова Т.С. Расовая принадлежность семян заразихи (Orobanche cumana Wallr.), собранных на полях разных регионов РФ в 2019 году / Т.С. Антонова, Н.М. Арасланова, Ю.В. Питинова // Аграрная наука. - 2020. - №6. - С. 62-65]. Затем, семена расы G заразихи вносятся в короба с почвенно-песчаной смесью одновременно с семенами подсолнечника. Растения подсолнечника выращиваются в течение 30 дней при температуре 25-27°С и 16-часовом фотопериоде. Через 30 дней после появления всходов растения выкапывают и проводят учет особей заразихи на их корнях. Восприимчивыми считаются растения, на корнях которых обнаружены клубеньки или сформировавшиеся побеги заразихи. Устойчивыми - растения, на корнях которых не было обнаружено здоровых клубеньков и побегов, но видны многочисленные некрозы клеток в области проникновения заразихи и погибшие клубеньки.
Этот способ обладает рядом недостатков, таких как: длительность процесса, трудозатратность и недостаточно высокая пропускная способность. В первую очередь это связано с тем, что необходимо создание инфекционного фона в теплице из семян расы G заразихи, для чего необходимо предварительно определить расовый состав семян заразихи.
Использование ДНК-маркеров, основанных на методе полимеразной цепной реакции (ПЦР), способных отличить различные аллели целевого гена, позволяет проводить отбор нужных генотипов без фитопатологической оценки на любой стадии развития подсолнечника, что может существенно сократить время, снизить трудозатраты, и повысить точность отбора растений подсолнечника, несущих ген устойчивости подсолнечника к расе G заразихи.
Разработка и дальнейшее использование маркерных систем для ускоренной селекции подсолнечника активно применяется во многих странах. Расоспецифичный ген, контролирующий устойчивость к расе Е - Or5, картирован в теломерной области хромосомы 3 на 7,5 сМ выше маркера ORS1036 [Tang S. Genetic mapping of the Or5 gene for resistance to Orobanche race E in sunflower / S. Tang, A. Heesacker, V.K. Kishore, A. Fernandez, E.S. Sadik, G. Cole, S.J. Knapp // Crop Science. - 2003. - T. 43. - №3. - C. 1021-1028]. В той же хромосоме позже был локализован ген устойчивости к расе выше F - orab-v1-8 [Imerovski I. Mapping of a new gene for resistance to broomrape races higher than F / I. Imerovski, A. Dimitrijević, D. Miladinović, B. Dedić, S. Jocić, N.K. Tubić, S. Cvejić // Euphytica. - 2016. -T. 209. - C. 281-289]. Также один ген устойчивости к расе F HaOr7 картирован на хромосоме 7 [Duriez Р.A receptor-like kinase enhances sunflower resistance to Orobanche cumana / P. Duriez, S. Vautrin, M.C. Auriac, J. Bazerque, M.C. Boniface, C. Callot, et al. // Nature Plants. - 2019. - Т. 5. - №12. - С.1211-1215]. По некоторым данным, этот ген также может контролировать устойчивость к расам выше F [Martín-Sanz A. Pérez-Vich В., Rueda S., Fernández-Martínez J. M., Velasco L. Characterization of post-haustorial resistance to sunflower broomrape / A. Martín-Sanz, B. Pérez-Vich, S. Rueda, J. M. Fernández-Martínez, L. Velasco // Crop Science. - 2020. - T. 60. - №3. - C. 1188-1198. https://doi.org/10.1002/csc2.20002].
Ген устойчивости к pace G OrDeb2, интрогрессированный в культурный подсолнечник от Н. debilis subsp.tardiflorus, картирован в верхней половине хромосомы 4 [Fernández-Aparicio М., Del Moral L., Muños S., Velasco L., Pérez-Vich B. Genetic and physiological characterization of sunflower resistance provided by the wild-derived OrDeb2 gene against highly virulent races of Orobanche cumana Wallr / M. Fernández-Aparicio, L. Del Moral, S. Muños, L. Velasco, B. Pérez-Vich // Theoretical and Applied Genetics. - 2022. - T. 135. - №. 2.-C. 501-525].
Для обнаружения этого гена в растениях подсолнечника компанией ДАУ АГРОСАЙЕНСИЗ ЛЛС (US) разработаны ДНК-маркеры ([http://new.fips.ru/registers-doc-view/fips_servlet?DB=RUPAT&DocNumber=2776361&TypeFile=html Патент №2776361, СПК А01Н1/04, А01Н6/14, С07Н21/00, C12Q1/68, опубл. 19.07.2022], «Молекулярные маркеры, ассоциированные с устойчивостью подсолнечника к Orobanche».
Главным недостатком данного способа отбора растения подсолнечника, устойчивого к заразихе (Orobanche), является то, что описанные ДНК-маркеры применимы для определения только тех устойчивых растений подсолнечника, которые несут ген OrDeb2 и непригодны, в случае, когда растение несет другой ген устойчивости. Также, данные ДНК-маркеры являются сцепленными с целевым геном, это означает, что они обладают определенной вероятностью ошибки определения, в отличии от внутригенных маркеров.
Сущность изобретения.
Задачей предлагаемого изобретения является разработка системы внутригенных ДНК-маркеров, которая позволит идентифицировать доминантный и рецессивный аллели гена HaOr7, описанного P. Duriez и др., в линиях, гибридах и селекционном материале подсолнечника на любых стадиях развития растений с использованием рутинных методов молекулярной биологии, в том числе метода полимеразной цепной реакции (ПЦР). Тот факт, что разработанная система дает возможность высокой точностью отличать устойчивые и восприимчивые растения, доказывает то, что все устойчивые растения подсолнечника, возделываемые в Российской Федерации, несут именно ген HaOr7, который, следовательно контролирует устойчивость к расе G заразихи.
Техническим результатом изобретения является возможность проведения отбора растений подсолнечника на устойчивость к расе G заразихи с помощью ПЦР, не прибегая к фитопатологической оценке, что значительно повышает эффективность и точность отбора.
Поставленная задача достигается предлагаемой системой из 2 ДНК-маркеров, представляющим собой 2 пары специфических олигонуклеотидных праймеров, позволяющих амплифицировать последовательность ДНК подсолнечника и выявлять доминантные и рецессивные аллели гена HaOr7 одновременно:
Для разработки указанных олигонуклеотидных праймеров было проведено выравнивание нуклеотидных последовательностей доминантного и рецессивного аллелей гена HaOr7, Or7-1 (номер в базе данных NCBI: MN219479.1) и LOC110867090 receptor kinase-like protein Xa21 из 7 хромосомы референсного генома подсолнечника HanXRQr2.0-SUNRISE (номер в базе данных NCBI: NC_035439.2 (143278893..143280718, complement)), соответственно, которые были найдены в базе данных нуклеотидных последовательностей NCBI (URL:https://www.ncbi.nlm.nih.gov/).
Специфические олигонуклеотидные праймеры были сконструированы таким образом, что пара праймеров RORS1_F и RORS1_R отжигалась только на последовательность Or7-1, а пара праймеров SORS9_F и SORS9_R только на последовательность референсного генома подсолнечника HanXRQr2.0-SUNRISE, а также чтобы ПЦР-продукты, получаемые с их помощью, отличались по длине между доминантным и рецессивным аллелями и составили 168 и 217 пар нуклеотидов, соответственно. Учитывались параметры температуры отжига и отсутствия образования вторичных структур между праймерами, что позволяет проводить мультиплексную ПЦР сразу с 2 парами праймерами.
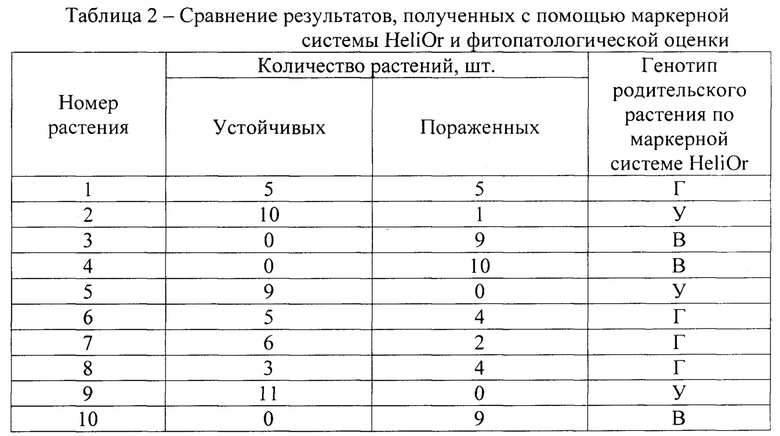
Разработанная система ДНК-маркеров была протестирована на 48 образцах различных по устойчивости к расе G заразихи линий и гибридов подсолнечника, что указано в таблице 1.
На фиг.1 представлен результат электрофоретического анализа амплификации геномной ДНК этих образцов с использованием разработанной системы ДНК-маркеров (HeliOr). Как видно на фиг.1, система ДНК-маркеров показала 100% соответствие наличия доминантного аллеля гена HaOr7 и устойчивости подсолнечника к расе G заразихи (образцы под номерами 1-15,), отсутствие доминантного аллеля гена HaOr7 и наличие рецессивного аллеля гена было выявлено у восприимчивых к заразихе линий подсолнечника (образцы под номерами 16-30), также оба аллеля были выявлены у гибридов подсолнечника, полученных от скрещиваний устойчивых и восприимчивых линий ВНИИМК, и коммерческих гибридов ВНИИМК и иностранных компаний (образцы под номерами 30-46).
Представленные данные указывают на то, что разработанная система ДНК-маркеров может быть использована для отбора растений подсолнечника, несущих ген устойчивости к расе G заразихи, а также определения наличия восприимчивого аллеля у устойчивых растений.
Перечень последовательностей олигонуклеотидных праймеров.
Первая пара праймеров:
SEQ ID NO 1 (RORS1_F): 5'-ACCATCTTTGCTTCCTATGGCA-3'
SEQ ID NO 2 (RORS1_R): 5'-CGTCACCCTTCTATGTCGCT-3'
Вторая пара праймеров:
SEQ ID NO 3 (SORS9_F): 5'-AGTGCATTCGCAATCTTCGC-3'
SEQ ID NO 4 (SORS9_R): 5'-TGCCATCCTCATCATTAAGGG-3'
Осуществление изобретения.
Пример 1
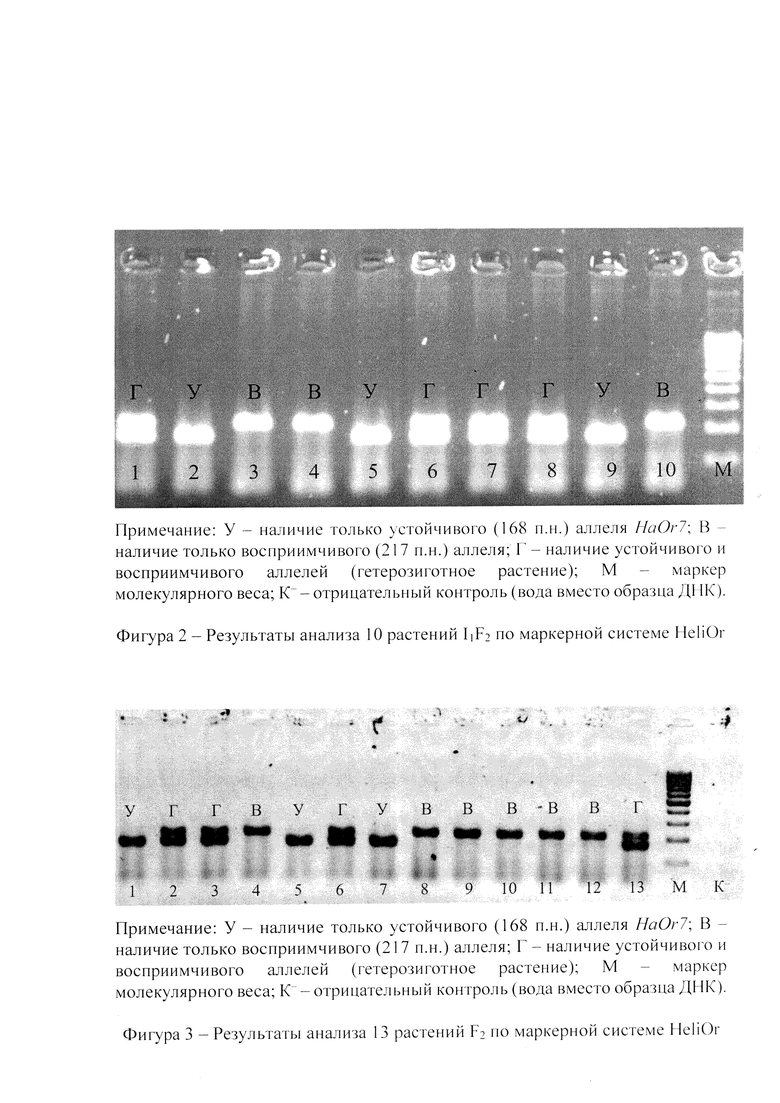
Предлагаемая система ДНК-маркеров, названная нами HeliOr была использована для оценки растений F2 при создании новых устойчивых к расе G заразихи линий подсолнечника после скрещивания устойчивой линии, несущей доминантный аллель гена HaOr7 с восприимчивой линией. Полученные после самоопыления растений поколения F1 семена F2 высаживали в почву. ДНК выделяли из молодых листьев всех высаженных растений. Для этого 2-3 кусочка листа (массой примерно 100 мг) помещали в пробирку Eppendorf объемом 1,5 мл. Экстракцию ДНК проводили методом с применением цетилтриметиламмония бромида (ЦТАБ). Оценку качества и количества экстрагированной ДНК производили путем проведения аналитического электрофореза. Выделенная ДНК использовалась в качестве матрицы в ПЦР, которую проводили в реакционной смеси объемом 25 мкл, содержащей 2 мкл экстрагированной ДНК, 200 мкМ дНТФ, по 10 пкМ каждого праймера, входящего в 2 праймерные пары системы ДНК-маркеров 2,5 мкл ПЦР-Буфера для Taq ДНК-полимеразы, 2,5 мМ MgCl2, 1 единица активности Taq ДНК-полимеразы. Постановку ПЦР проводили в амплификаторе по следующей программе: 5 мин. при 94°С для начальной денатурации; следующие 35 циклов: денатурация 40 сек. при 94°С; 30 сек. отжиг праймеров при температуре 60°С; 40 сек. синтез при 72°С. Финальный цикл синтеза при 72°С - 5 мин. Результаты электрофоретического анализа продуктов ПЦР в 2% агарозном геле представлены на фиг.2. Растениям был присвоены генотипы: У - наличие только устойчивого (168 п.н.) аллеля HaOr7; В - наличие только восприимчивого (217 п.н.) аллеля; Г - наличие устойчивого и восприимчивого аллелей (гетерозиготное растение). Таким образом, установлены следующие генотипы образцов: устойчивые гомозиготные растения под номерами 2, 5, 9; восприимчивые гомозиготные растения под номерами 3, 4, 10 и гетерозиготные растения под номерами 1, 6-8. Полученные результаты были переданы в лабораторию генетики отдела селекции и первичного семеноводства подсолнечника ФГБНУ ФНЦ ВНИИМК (Краснодар). Все растения F2, оцененные с помощью маркерной системы, были самоопылены. Далее была проведена фитопатологическая оценка потомства - растений IiF2 и подтверждено полное совпадение результатов (таблица 2).

Пример 2.
Разработанная система ДНК-маркеров была использована для отбора растений в поколении F2 от скрещивания линии ВК680 и линии донора устойчивости к расе G заразихи RGM. В результате ПЦР-анализа геномной ДНК с использованием системы ДНК-маркеров HeliOr, из 13 растений были выявлены три растения, у которых на электрофореграмме присутствовал единичный ПЦР-продукт длиной 168 пар нуклеотидов, соответствующий доминантному аллелю HaOr7 (устойчивые гомозиготные растения) (фиг. 3). Также были отобраны гетерозиготные растения, несущие доминантный аллель HaOr7. Отобранные растения были самоопылены и потомство было оценено на зараженной семенами расы G заразихи почве. Все растения потомства гомозиготных растений показали полную устойчивость к расе G заразихи. Потомство гетерозиготных растений было высажено в почву. Среди них помощью маркерной системы были отобраны и самоопылены только гомозиготные растения, несущие только доминантный аллель HaOr7.
Таким образом, разработанная система ДНК-маркеров (HeliOr) может быть применена для отбора растений подсолнечника, устойчивого к расе G заразихи. Использование предлагаемой системы ДНК-маркеров позволит существенно снизить трудозатраты, повысить точность и скорость отбора по сравнению с отбором по фенотипу с помощью фитопатологической оценки или с помощью сцепленных с целевым геном ДНК-маркеров.
Список литературы:
1. Антонова Т.С. Заразиха на подсолнечнике. - Краснодар: Просвещение-Юг, 2018. - 58 с.
2. Лукомец В.М. Современные тренды селекционно-генетического улучшения сортов и гибридов подсолнечника во ВНИИМК / В.М. Лукомец, М.В. Трунова, Я.Н. Демурин // Вавиловский журнал генетики и селекции. - 2021. - Т. 25. - №4. - С.388-393
3. Антонова Т.С. Отбор на устойчивость к расе G заразихи из расщепляющихся популяций подсолнечника в искусственных условиях выращивания / Т.С. Антонова, Е.А. Стрельников, Н.М. Арасланова, С.З. Гучетль, Т.А. Челюстникова // Масличные культуры. Научно-технический бюллетень Всероссийского научно-исследовательского института масличных культур. -2017. - №3(171). - С.18-22
4. Антонова Т.С. Расовая принадлежность семян заразихи (Orobanche cumana Wallr.), собранных на полях разных регионов РФ в 2019 году / Т.С. Антонова, Н.М. Арасланова, Ю.В. Питинова // Аграрная наука. - 2020. - №6. - С. 62-65
5. Tang S., Heesacker A., Kishore V.K., Fernandez A., Sadik Е.S., Cole G., Knapp S.J. Genetic mapping of the Or5 gene for resistance to Orobanche race E in sunflower / S. Tang, A. Heesacker, V.K. Kishore, A. Fernandez, E.S. Sadik, G. Cole, S.J. Knapp // Crop Science. - 2003. - T. 43. - №3. - С. 1021-1028
6. Imerovski I. Mapping of a new gene for resistance to broomrape races higher than F /I. Imerovski, A. Dimitrijević, D. Miladinović, B. Dedić, S. Jocić, N. K. Tubić, S. Cvejić // Euphytica. - 2016. - T. 209. - C. 281-289
7. Duriez P. A receptor-like kinase enhances sunflower resistance to Orobanche cumana / P. Duriez, S. Vautrin, M. C. Auriac, J. Bazerque, M. C. Boniface, C. Callot, et al. // Nature Plants. - 2019. - T. 5. - №12. - C. 1211-1215.
8. Martín-Sanz A. Pérez-Vich В., Rueda S., Fernández-Martínez J. M., Velasco L. Characterization of post-haustorial resistance to sunflower broomrape / A. Martín-Sanz, B. Pérez-Vich, S. Rueda, J. M. Fernández-Martínez, L. Velasco // Crop Science. - 2020. - T. 60. - №3. - C. 1188-1198. https://doi.org/10.1002/csc2.20002.
9. Fernández-Aparicio M., Del Moral L., Muños S., Velasco L., Pérez-Vich B. Genetic and physiological characterization of sunflower resistance provided by the wild-derived OrDeb2 gene against highly virulent races of Orobanche cumana Wallr / M. Fernández-Aparicio, L. Del Moral, S. Muños, L. Velasco, B. Pérez-Vich // Theoretical and Applied Genetics. - 2022. - T. 135. - №. 2.- C. 501-525.
10. http://new.fips.ru/registers-doc-view/fips_servlet?DB=RUPAT&DocNumber=2776361&TypeFile=html
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="ru"
dtdVersion="V1_3" fileName="Система внутригенных ДНК-маркеров для
детекции в растениях подсолнечника доминантного и рецессивного
аллелей гена HaOr7, контролирующего устойчивость к расе G заразихи
(Orobanche cumana Wallr).xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2023-08-11">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1</ApplicationNumberText>
<FilingDate></FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>1</ApplicantFileReference>
<ApplicantName languageCode="ru"> Федеральное государственное
бюджетное научное учреждение «Федеральный научный центр
«Всероссийский научно-исследовательский институт масличных культур
имени В.С. Пустовойта»</ApplicantName>
<ApplicantNameLatin>V.S. Pustovoit All-Russian Research Institute of
Oil Crops</ApplicantNameLatin>
<InventorName languageCode="ru">Савиченко Дмитрий
Леонидович</InventorName>
<InventorNameLatin>Savichenko Dmitrii
Leonidovich</InventorNameLatin>
<InventionTitle languageCode="ru">Система внутригенных ДНК-маркеров
для детекции в растениях подсолнечника доминантного и рецессивного
аллелей гена HaOr7, контролирующего устойчивость к расе G заразихи
(Orobanche cumana Wallr.).</InventionTitle>
<SequenceTotalQuantity>4</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q3">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>accatctttgcttcctatggca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtcacccttctatgtcgct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q7">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agtgcattcgcaatcttcgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q9">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgccatcctcatcattaaggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ определения устойчивости растений подсолнечника однолетнего к заразихе кумской расы G | 2023 |
|
RU2823806C1 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ, АССОЦИИРОВАННЫЕ С УСТОЙЧИВОСТЬЮ ПОДСОЛНЕЧНИКА К OROBANCHE | 2018 |
|
RU2776361C2 |
| ПЛАТФОРМА ДЛЯ ФЕНОТИПИРОВАНИЯ БИОЛОГИЧЕСКИХ ВОЗБУДИТЕЛЕЙ РАСТЕНИЙ И СПОСОБ ФЕНОТИПИРОВАНИЯ | 2014 |
|
RU2663026C2 |
| УСТОЙЧИВЫЕ К ГЕРБИЦИДАМ РАСТЕНИЯ ПОДСОЛНЕЧНИКА | 2009 |
|
RU2551781C2 |
| СПОСОБ ИДЕНТИФИКАЦИИ СТЕРИЛЬНОСТИ/ФЕРТИЛЬНОСТИ ПОДСОЛНЕЧНИКА | 2012 |
|
RU2524135C2 |
| Способ отбора линий яровой мягкой пшеницы с повышенным содержанием антоцианов в зерне | 2021 |
|
RU2762804C1 |
| Внутригенный ДНК-маркер для отбора пшеницы с повышенным содержанием антоцианов в перикарпе зерновки | 2021 |
|
RU2774444C1 |
| МОЛЕКУЛЯРНЫЙ МАРКЕР FR_ER1 И ЕГО ИСПОЛЬЗОВАНИЕ ДЛЯ СЕЛЕКЦИИ НОВЫХ СОРТОВ ГОРОХА, УСТОЙЧИВЫХ К МУЧНИСТОЙ РОСЕ | 2013 |
|
RU2593691C2 |
| Набор ДНК-маркеров для отбора ячменя с повышенным содержанием антоцианов в перикарпе зерновки | 2022 |
|
RU2797678C1 |
| СПОСОБ ОПРЕДЕЛЕНИЯ ВСХОЖЕСТИ СЕМЯН ЗАРАЗИХИ ПОДСОЛНЕЧНОЙ | 2010 |
|
RU2442318C2 |
Изобретение относится к сельскому хозяйству и биотехнологии, в частности к системе ДНК-маркеров для обнаружения в растениях подсолнечника доминантного и рецессивного аллелей гена HaOr7, контролирующего устойчивость к расе G заразихи Orobanche cumana Wallr. Изобретение позволяет повысить точность и эффективность селекционного процесса на устойчивость подсолнечника к расе G заразихи. 3 ил., 2 табл., 2 пр.
Система внутригенных ДНК-маркеров для обнаружения в растениях подсолнечника доминантного и рецессивного аллелей гена НаОr7, контролирующего устойчивость к расе G заразихи (Orobanche cumana Wallr.), представляющая собой 2 пары специфических олигонуклеотидных праймеров для полимеразной цепной реакции, указанных как SEQ ID NO: 1 и SEQ ID NO: 2 - первая пара, SEQ ID NO: 3 и SEQ ID NO: 4 - вторая пара, позволяющие амплифицировать специфичные последовательности ДНК подсолнечника, характеризующие доминантный и рецессивный аллели гена НаОr7 одновременно в одной мультиплексной ПЦР.
| DURIEZ P., et al., A receptor-like kinase enhances sunflower resistance to Orobanche Cumana, Nature Plants, 2019, V.5, pp.1211-1215 | |||
| WO 2011068261, 09.06.2011 | |||
| СПОСОБ ИДЕНТИФИКАЦИИ СТЕРИЛЬНОСТИ/ФЕРТИЛЬНОСТИ ПОДСОЛНЕЧНИКА | 2012 |
|
RU2524135C2 |
Авторы
Даты
2024-11-27—Публикация
2023-08-28—Подача