Область техники
Настоящее изобретение относится к методике манипулирования нуклеиновой кислотой с использованием белка, способного связываться с нуклеиновой кислотой-мишенью. Настоящее изобретение полезно в широком диапазоне областей, включая медицину (поддержка открытия лекарств, терапевтическое лечение и т.д.), сельское хозяйство (сельское хозяйство, рыболовство и животноводство, разведение и т.д.) и химию (производство биологических материалов и т.д.).
Уровень техники
Белки PPR представляют собой белки, содержащие повторы мотивов PPR, каждый из которых имеет длину около 35 аминокислот, и один мотив PPR может специфически связываться с одним основанием. Комбинация первой, четвертой и ii (второй с конца перед следующим мотивом) аминокислот в мотиве PPR определяет, с каким из аденина, цитозина, гуанина и урацила (или тимина) связывается мотив (Патентные документы 1 и 2).
Поскольку мотивы PPR достигают связывания путем распознавания одного основания с одним мотивом, при конструировании, например, белка PPR, который специфически связывается с последовательностью нуклеиновой кислоты длиной 18 оснований, 18 мотивов PPR должны быть связаны вместе. До сих пор сообщалось об искусственных белках PPR, содержащих от 7 до 14 мотивов PPR, связанных вместе (не патентные документы с 1 по 6).
Ссылки предшествующего уровня техники
Патентные документы
Патентный документ 1: международная публикация WO2013/058404.
Патентный документ 2: международная публикация WO2014/175284.
Не патентные документы
Не патентный документ 1: Coquille, S. et al., An artificial PPR scaffold for programmable RNA recognition, Nature Communications 5, Article number: 5729 (2014)
Не патентный документ 2: Shen, C. et al., Specific RNA Recognition by Designer Pentatricopeptide Repeat Protein, Molecular Plant 8, 667-670 (2015)
Не патентный документ 3: Shen, C. et al., Structural basis for specific single-stranded RNA recognition by designer pentatricopeptide repeat proteins, Nature Communications, Volume 7, Article number: 11285 (2016)
Не патентный документ 4: Gully, B.S. et al., The design and structural characterization of a synthetic pentatricopeptide repeat protein, Acta Cryst., D71, 196-208 (2015)
Не патентный документ 5: Miranda, R.G. et al., RNA-binding specificity landscapes of designer pentatricopeptide repeat proteins elucidate principles of PPR-RNA interactions, Nucleic Acids Research, 46(5), 2613-2623 (2018)
Не патентный документ 6: Yan, J. et al., Delineation of pentatricopeptide repeat codes for target RNA prediction, Nucleic Acids Research, gkz075 (2019)
Сущность изобретения
Задача, которую должно решить изобретение
Высокоэффективные белки PPR необходимы для того, чтобы белки PPR специфически связывались с молекулой РНК-мишени в клетках, и чтобы с ними можно было проводить желаемые манипуляции.
Кроме того, для того, чтобы белки PPR специфически связывались с молекулой РНК-мишени в клетках и чтобы с ними можно было проводить какие-либо желаемые манипуляции, необходимы белки PPR, которые содержат связанные мотивы в количестве более 7-14, которые применяют обычно, и могут связываться с более длинными последовательностями. Например, геномы человека содержат 6 миллиардов пар оснований, состоящих из четырех типов оснований (A, C, G и T или U), и поэтому требуется последовательность из, по меньшей мере, 17 нуклеотидов, чтобы определить одну нуклеотидную последовательность из последовательности геномов (это потому, что 416 равно 4 миллиардов, и 417 равно 16 миллиардов).
Средства для достижения цели
Настоящее изобретение представляет следующие элементы как новые мотивы PPR и так далее.
[1] Мотив PPR, который является одним из следующих мотивов PPR:
(A-1) мотив PPR, состоящий из последовательности SEQ ID NO: 9, или мотив PPR, состоящий из последовательности SEQ ID NO: 9, имеющий замену, выбранную из группы, состоящей из аминокислотной замены в положении 10 тирозином, аминокислотной замены в положении 15 лизином, аминокислотной замены в положении 16 лейцином, аминокислотной замены в положении 17 глутаминовой кислотой, аминокислотной замены в положении 18 аспарагиновой кислотой и аминокислотной замены в положении 28 глутаминовой кислотой, или мотив PPR, состоящий из последовательности SEQ ID NO: 401, или мотив PPR, состоящий из последовательности SEQ ID NO: 401, имеющей замену, выбранную из группы, состоящей из аминокислотной замены в положении 10 тирозином, аминокислотной замены в положении 16 лейцином, аминокислотной замены в положении 17 глутаминовой кислотой, аминокислотной замены в положении 18 аспарагиновой кислотой и аминокислотной замены в положении 28 глутаминовой кислотой;
(A-2) мотив PPR, состоящий из последовательности SEQ ID NO: 9 или 401, имеющей замену, делецию или добавление от 1 до 20 аминокислот, кроме аминокислот в положениях 1, 2, 3, 4, 6, 7, 9, 11, 12, 14, 19, 26, 30, 33 и 34, и обладающий свойством связывания аденина;
(A-3) мотив PPR, имеющий идентичность последовательности, по меньшей мере, 42% с последовательностью SEQ ID NO: 9 или 401, при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 7, 9, 11, 12, 14, 19, 26, 30, 33 и 34 идентичны, и обладающий свойством связывания аденина;
(C-1) мотив PPR, состоящий из последовательности SEQ ID NO: 10, или мотив PPR, состоящий из последовательности SEQ ID NO: 10, имеющей замену аминокислоты, выбранной из группы, состоящей из аминокислотной замены в положении 2 серином, аминокислотной замены в положении 5 изолейцином, аминокислотной замены в положении 7 лейцином, аминокислотной замены в положении 8 лизином, аминокислотной замены в положении 10 фенилаланином или тирозином, аминокислотной замены в положении 15 аргинином, аминокислотной замены в положении 22 валином, аминокислотной замены в положении 24 аргинином, аминокислотной замены в положении 27 лейцином и аминокислотной замены в положении 29 аргинином;
(C-2) мотив PPR, состоящий из последовательности SEQ ID NO: 10, имеющей замену, делецию или добавление от 1 до 25 аминокислот, кроме аминокислот в положениях 1, 3, 4, 14, 18, 19, 26, 30, 33 и 34, и обладающий свойством связывания цитозина;
(C-3) мотив PPR, имеющий идентичность последовательности, по меньшей мере, на 25% с последовательностью SEQ ID NO: 10, при условии, что аминокислоты в положениях 1, 3, 4, 14, 18, 19, 26, 30, 33 и 34 идентичны, и обладающий свойством связывания цитозина;
(G-1) мотив PPR, состоящий из последовательности SEQ ID NO: 11, или мотив PPR, состоящий из последовательности SEQ ID NO: 11, имеющий замену, выбранную из группы, состоящей из аминокислотной замены в положении 10 фенилаланином, аминокислотной замены в положении 15 аспарагиновой кислотой, аминокислотной замены в положении 27 валином, аминокислотной замены в положении 28 серином и аминокислотной замены в положении 35 изолейцином;
(G-2) мотив PPR, состоящий из последовательности SEQ ID NO: 11, имеющей замену, делецию или добавление от 1 до 21 аминокислот, кроме аминокислот в положениях 1, 2, 3, 4, 6, 7, 9, 14, 18, 19, 26, 30, 33 и 34, и обладающий свойством связывания гуанина;
(G-3) мотив PPR, имеющий идентичность последовательности, по меньшей мере, 40% с последовательностью SEQ ID NO: 11, при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 7, 9, 14, 18, 19, 26, 30, 33 и 34 идентичны, и обладающий свойством связывания гуанина;
(U-1) мотив PPR, состоящий из последовательности SEQ ID NO: 12, или мотив PPR, состоящий из последовательности SEQ ID NO: 12, имеющий замену, выбранную из группы, состоящей из аминокислотной замены в положении 10 фенилаланином, аминокислотной замены в положении 13 серином, аминокислотной замены в положении 15 лизином, аминокислотной замены в положении 17 глутаминовой кислотой, аминокислотной замены в положении 20 лейцином, аминокислотной замены в положении 21 лизином, аминокислотной замены в положении 23 фенилаланином, аминокислотной замены в положении 24 аспарагиновой кислотой, аминокислотной замены в положении 27 лизином, аминокислотной замены в положении 28 лизином, аминокислотной замены в положении 29 аргинином и аминокислотной замены в положении 31 лейцином;
(U-2) мотив PPR, состоящий из последовательности SEQ ID NO: 12, имеющей замену, делецию или добавление от 1 до 22 аминокислот, кроме аминокислот в положениях 1, 2, 3, 4, 6, 11, 12, 14, 19, 26, 30, 33 и 34, и обладающий свойством связывания урацила; и
(U-3) мотив PPR, имеющий идентичность последовательности, по меньшей мере, 37% с последовательностью SEQ ID NO: 12, при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 11, 12, 14, 19, 26, 30, 33 и 34 идентичны, и обладающий свойством связывания урацила.
[2] Использование мотива PPR по 1 для получения белка PPR, РНК-мишень которого имеет длину 15 оснований или более.
[3] Использование мотива PPR по 1 для получения белка PPR, который предназначен для повышения эффективности связывания белка PPR с РНК-мишенью.
[4] Белок PPR, содержащий n мотивов PPR и способный связываться с РНК-мишенью, состоящей из последовательности из n оснований длиной, где:
мотивом PPR для аденина в последовательности оснований является мотив PPR (A-1), (A-2) или (A-3), определенный в 1;
мотивом PPR для цитозина в последовательности оснований является мотив PPR (C-1), (C-2) или (С-3), определенный в 1;
мотивом PPR для гуанина в последовательности оснований является мотив PPR (G-1), (G-2) или (G-3), определенный в 1; и
мотивом PPR для урацила в последовательности оснований является мотив PPR (U-1), (U-2) или (U-3), определенный в 1.
[5] Белок по 4, где n равно 15 или больше.
[6] Белок по 4 или 5, где первым мотивом PPR от N-конца является любой из следующих мотивов:
(1st_A-1) мотив PPR, состоящий из последовательности SEQ ID NO: 402, имеющей такие аминокислотные замены в положениях 6 и 9, которые удовлетворяет любая из комбинаций, определенных ниже;
(1st_A-2) мотив PPR, состоящий из последовательности (1st_A-1), имеющей замену, делецию или добавление от 1 до 9 аминокислот, кроме аминокислот в положениях 1, 4, 6, 9 и 34, и обладающий свойством связывания аденина;
(1st_A-3) мотив PPR, имеющий идентичность последовательности, по меньшей мере, 80% с последовательностью (1st_A-1), при условии, что аминокислоты в положениях 1, 4, 6, 9 и 34 идентичны, и обладающий свойство связывания аденина;
(1st_С-1) мотив PPR, состоящий из последовательности SEQ ID NO: 403;
(1st_C-2) мотив PPR, содержащий последовательность (1st_C-1), имеющую замену, делецию или добавление от 1 до 9 аминокислот, кроме аминокислот в положениях 1, 4, 6, 9 и 34, и обладающий свойством связывания цитозина;
(1st_C-3) мотив PPR, имеющий идентичность последовательности, по меньшей мере, 80% с последовательностью (1st_C-1), при условии, что аминокислоты в положениях 1, 4, 6, 9 и 34 идентичны, и обладающий свойством связывания цитозина;
(1-й_G-1) мотив PPR, состоящий из последовательности SEQ ID NO: 404, имеющей такие аминокислотные замены в положениях 6 и 9, которые удовлетворяет любая из комбинаций, определенных ниже;
(1st_G-2) мотив PPR, содержащий последовательность (1st_G-1), имеющую замену, делецию или добавление от 1 до 9 аминокислот, кроме аминокислот в положениях 1, 4, 6, 9 и 34, и обладающий свойством связывания гуанина;
(1st_G-3) мотив PPR, имеющий идентичность последовательности, по меньшей мере, 80% с последовательностью (1st_G-1), при условии, что аминокислоты в положениях 1, 4, 6, 9 и 34 идентичны, и обладающий свойством связывания гуанина;
(1-й_U-1) мотив PPR, состоящий из последовательности SEQ ID NO: 405, имеющей такие аминокислотные замены в положениях 6 и 9, которые удовлетворяет любая из комбинаций, определенных ниже;
(1st_U-2) мотив PPR, содержащий последовательность (1st_U-1), имеющую замену, делецию или добавление от 1 до 9 аминокислот, кроме аминокислот в положениях 1, 4, 6, 9 и 34, и обладающий свойством связывания урацила; и
(1st_U-3) мотив PPR, имеющий идентичность последовательности, по меньшей мере, 80% с последовательностью (1st_U-1), при условии, что аминокислоты в положениях 1, 4, 6, 9 и 34 идентичны, и обладающий свойством связывания урацила:
- комбинация аспарагина в качестве аминокислоты в положении 6 и глутаминовой кислоты в качестве аминокислоты в положении 9,
- комбинация аспарагина в качестве аминокислоты в положении 6 и глутамина в качестве аминокислоты в положении 9,
- комбинация аспарагина в качестве аминокислоты в положении 6 и лизина в качестве аминокислоты в положении 9, и
- комбинация аспарагиновой кислоты в качестве аминокислоты в положении 6 и глицина в качестве аминокислоты в положении 9.
[7] Способ контроля сплайсинга РНК, в котором используют белок по любому из 4-6.
[8] Способ определения РНК, в котором используют белок по любому из 4-6.
[9] Слитый белок, по меньшей мере, одного, выбранного из группы, состоящей из флуоресцентного белка, сигнального пептида ядерной локализации и белка-метки, и белка по любому из 4-6.
[10] Нуклеиновая кислота, кодирующая мотив PPR по 1 или белок по любому из 4-6.
[11] Вектор, содержащий нуклеиновую кислоту по 10.
[12] Клетка (за исключением человека), содержащая вектор по 11.
[13] Способ манипулирования РНК, в котором используют мотив PPR по 1, белок по любому из 4-6 или вектор по 11 (реализация на человеке исключена).
[14] Способ получения организма, включающий способ манипуляции по 13.
[1] Мотив PPR, которым является одним из следующих мотивов PPR:
(A-1) мотив PPR, состоящий из последовательности SEQ ID NO: 9, или мотив PPR, состоящий из последовательности SEQ ID NO: 9, имеющий замену, выбранную из группы, состоящей из аминокислотной замены в положении 10 тирозином, аминокислотной замены в положении 15 лизином, аминокислотной замены в положении 16 лейцином, аминокислотной замены в положении 17 глутаминовой кислотой, аминокислотной замены в положении 18 аспарагиновой кислотой и аминокислотной замены в положении 28 глутаминовой кислотой;
(A-2) мотив PPR, состоящий из последовательности SEQ ID NO: 9, имеющей замену, делецию или добавление от 1 до 20 аминокислот, кроме аминокислот в положениях 1, 2, 3, 4, 6, 7, 9, 11, 12, 14, 19, 26, 30, 33 и 34, и обладающий свойством связывания аденина;
(A-3) мотив PPR, имеющий идентичность последовательности, по меньшей мере, 42% с последовательностью SEQ ID NO: 9, при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 7, 9, 11, 12, 14, 19, 26, 30, 33 и 34 идентичны, и обладающий свойством связывания аденина;
(C-1) мотив PPR, состоящий из последовательности SEQ ID NO: 10, или мотив PPR, состоящий из последовательности SEQ ID NO: 10, имеющей замену аминокислоты, выбранной из группы, состоящей из аминокислотной замены в положении 2 серином, аминокислотной замены в положении 5 изолейцином, аминокислотной замены в положении 7 лейцином, аминокислотной замены в положении 8 лизином, аминокислотной замены в положении 10 фенилаланином или тирозином, аминокислотной замены в положении 15 аргинином, аминокислотной замены в положении 22 валином, аминокислотной замены в положении 24 аргинином, аминокислотной замены в положении 27 лейцином и аминокислотной замены в положении 29 аргинином;
(C-2) мотив PPR, состоящий из последовательности SEQ ID NO: 10, имеющей замену, делецию или добавление от 1 до 25 аминокислот, кроме аминокислот в положениях 1, 3, 4, 14, 18, 19, 26, 30, 33 и 34, и обладающий свойством связывания цитозина;
(C-3) мотив PPR, имеющий идентичность последовательности, по меньшей мере, 25% с последовательностью SEQ ID NO: 10, при условии, что аминокислоты в положениях 1, 3, 4, 14, 18, 19, 26, 30, 33 и 34 идентичны, и обладающий свойством связывания цитозина;
(G-1) мотив PPR, состоящий из последовательности SEQ ID NO: 11, или мотив PPR, состоящий из последовательности SEQ ID NO: 11, имеющий замену, выбранную из группы, состоящей из аминокислотной замены в положении 10 фенилаланином, аминокислотной замены в положении 15 аспарагиновой кислотой, аминокислотной замены в положении 27 валином, аминокислотной замены в положении 28 серином и аминокислотной замены в положении 35 изолейцином;
(G-2) мотив PPR, состоящий из последовательности SEQ ID NO: 11, имеющей замену, делецию или добавление от 1 до 21 аминокислот, кроме аминокислот в положениях 1, 2, 3, 4, 6, 7, 9, 14, 18, 19, 26, 30, 33 и 34, и обладающий свойством связывания гуанина;
(G-3) мотив PPR, имеющий идентичность последовательности, по меньшей мере, 40% с последовательностью SEQ ID NO: 11, при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 7, 9, 14, 18, 19, 26, 30, 33 и 34 идентичны, и обладающий свойством связывания гуанина;
(U-1) мотив PPR, состоящий из последовательности SEQ ID NO: 12, или мотив PPR, состоящий из последовательности SEQ ID NO: 12, имеющий замену, выбранную из группы, состоящей из аминокислотной замены в положении 10 фенилаланином, аминокислотной замены в положении 13 серином, аминокислотной замены в положении 15 лизином, аминокислотной замены в положении 17 глутаминовой кислотой, аминокислотной замены в положении 20 лейцином, аминокислотной замены в положении 21 лизином, аминокислотной замены в положении 23 фенилаланином, аминокислотной замены в положении 24 аспарагиновой кислотой, аминокислотной замены в положении 27 лизином, аминокислотной замены в положении 28 лизином, аминокислотной замены в положении 29 аргинином и аминокислотной замены в положении 31 лейцином;
(U-2) мотив PPR, состоящий из последовательности SEQ ID NO: 12, имеющей замену, делецию или добавление от 1 до 22 аминокислот, кроме аминокислот в положениях 1, 2, 3, 4, 6, 11, 12, 14, 19, 26, 30, 33 и 34, и обладающий свойством связывания урацила; и
(U-3) мотив PPR, имеющий идентичность последовательности, по меньшей мере, 37% с последовательностью SEQ ID NO: 12, при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 11, 12, 14, 19, 26, 30, 33 и 34 идентичны, и обладающий свойством связывания урацила.
[2] Использование мотива PPR по 1 для получения белка PPR, РНК-мишень которого имеет длину 15 оснований или более.
[3] Использование мотива PPR по 1 для получения белка PPR, который предназначен для повышения эффективности связывания белка PPR с РНК-мишенью.
[4] Белок, содержащий n мотивов PPR и способный связываться с РНК-мишенью, состоящей из последовательности из n оснований длиной, где:
мотивом PPR для аденина в последовательности оснований является мотив PPR (A-1), (A-2) или (A-3), определенный в 1;
мотивом PPR для цитозина в последовательности оснований является мотив PPR (C-1), (C-2) или (С-3), определенный в 1;
мотивом PPR для гуанина в последовательности оснований является мотив PPR (G-1), (G-2) или (G-3), определенный в 1; и
мотивом PPR для урацила в последовательности оснований является мотив PPR (U-1), (U-2) или (U-3), определенный в 1.
[5] Белок по 4, где n равно 15 или больше.
[6] Способ контроля сплайсинга РНК, в котором используют белок по 4 или 5.
[7] Способ обнаружения РНК, в котором используют белок по 4 или 5.
[8] Слитый белок, по меньшей мере, одного, выбранного из группы, состоящей из флуоресцентного белка, сигнального пептида ядерной локализации и белка-метки, и белка по 4 или 5.
[9] Нуклеиновая кислота, кодирующая мотив PPR по 1 или белок по 4 или 5.
[10] Вектор, содержащий нуклеиновую кислоту по 9.
[11] Клетка (за исключением человека), содержащая вектор по 10.
[12] Способ манипулирования РНК, в котором используют мотив PPR по пункту 1, белок по пункту 4 или 5 или вектор по пункту 10 (реализация у человека исключена).
[13] Способ получения организма, включающий способ манипуляции по 12.
[14] Способ получения гена, кодирующего белок, содержащий n мотивов PPR, которые могут связываться с нуклеиновой кислотой-мишенью, состоящей из последовательности из n оснований длиной, который включает следующие стадии:
выбор m видов частей PPR, необходимых для получения целевого гена, из библиотеки, состоящей, по меньшей мере, из 20 x m видов частей PPR, которые состоят по меньшей мере, из m видов промежуточных векторов Dest-a, ..., которые сконструированы так, чтобы они могут последовательно связываться, и в каждый из них вставлено, по меньшей мере, 20 видов полинуклеотидов, включая 4 вида, кодирующие мотивы PPR, которые обладают свойствами связывания аденина, цитозина, гуанина и урацила или тимина, соответственно, и 16 видов, кодирующих продукты связывания двух из мотивов PPR, соответственно; и
реакция Golden Gate выбранных m видов частей PPR вместе с частями вектора для получения вектора, в который вставлено m продуктов полинуклеотидного связывания (где n равно m или больше, и равно m x 2 или меньше).
[15] Способ получения по 14, где m равно 10, и который предназначен для получения гена, кодирующего белок, содержащий 15 или более мотивов PPR.
[16] Способ определения или количественной оценки белка, содержащего n мотивов PPR, которые могут связываться с нуклеиновой кислотой-мишенью, состоящей из последовательности из n оснований длиной, который включает следующую стадию:
стадия добавления раствора, содержащего кандидатный белок, к твердофазной нуклеиновой кислоте-мишени, и определения или количественного определения белка, связавшегося с нуклеиновой кислотой-мишенью.
[17] Способ по п.16, в котором кандидатный белок сливают с маркерным белком.
Краткое описание чертежей
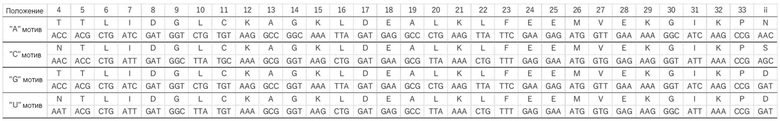
[Фиг. 1] v2 Мотив (распознающий аденин)
[Фиг. 2] v2 Мотив (распознающий цитозин)
[Фиг. 3] v2 Мотив (распознающий гуанин)
[Фиг. 4] v2 Мотив (распознающий урацил)
[Фиг. 5] Пример способа клонирования для бесшовного связывания последовательностей мотивов PPR.
[Фиг. 6] Проверка бесшовного клонирования с использованием библиотек мотивов PPR, содержащих 1 или 2 мотива. A: Аминокислотные последовательности мотивов v1, v2, v3.1 и v3.2. v3.1 соответствует v2, введенному с мутацией D15K в мотиве распознавания аденина. Для мотива v3.2 для первого мотива выбирается 1st_x, а второй и последующие мотивы выбираются из v2_C, v2_G, v2_U и v3.1_A. B: Результаты получения трех клонов для каждого из трех типов 18-мотивных белков PPR. С v1 были получены полосы правильного размера, за исключением второго клона PPR2. С v2 для всех клонов были получены полосы правильного размера.
[Фиг. 7] Пример высокопроизводительной оценки эффективности связывания РНК-связывающих белков. A: Сравнение типовых схем экспериментов по связыванию нуклеиновой кислоты с белком. B: Схема RPB-ELISA (ELISA связывания РНК-белка). C: Экспериментальные результаты, полученные с белком MS2 и его РНК-мишенью. Специфические связывания были определены как с раствором очищенного белка (очищенный белок), так и с лизатом E. coli (лизат).
[Фиг. 8] Результаты сравнительных экспериментов по связыванию РНК. Когда белки получают с последовательностью мотива v2, наблюдают увеличение (от 1,3 до 3,6 раза) способности связывания с последовательностью-мишенью для всех белков по сравнению с белками, полученными с последовательностью мотива v1. Кроме того, более высокий сигнал связывания с мишенью/сигнал связывания вне мишени (S/N) был получен с v2 для всех белков по сравнению с белками, полученными с помощью v1, что указывает на то, что v2 обеспечивает более высокую аффинность и специфичность для мишени по сравнению с v1. На верхнем левом графике (сигнал связывания (LU/107 CPS)) черные столбцы показывают результаты для зондов РНК, имеющих последовательности-мишени, серые столбцы показывают результаты для зондов РНК, имеющих 1 вне мишени, а белые столбцы показывают результаты для РНК. зонды, имеющие 2 вне мишени. В последовательностях зондов (Посл. зонда), показанных в нижней левой части, подчеркивание указывает на последовательности-мишени (Посл. мишень).
[Фиг. 9] Подробный анализ характеристик связывания РНК (специфичности) белков PPR. Белки PPR для 23 видов последовательностей-мишеней получают с использованием мотива v2, и все комбинации связывания анализируют с помощью RPB-ELISA. Было определено, что 21 вид белков PPR показал самую сильную связывающую способность со своими мишенями (верхняя часть). Аналогичным образом эффективность связывания РНК анализируют с использованием мотива V3.1 (нижняя часть).
[Фиг. 10] Подробный анализ характеристик связывания РНК (аффинности) белков PPR. A: Значения Kd полученных белков для их мишеней. Минимальное значение составляет 1,95×10-9, что является самым низким значением Kd среди полученных белков PPR, о которых сообщалось до сих пор. B: Корреляция между значением Kd и значением сигнала, полученным в эксперименте по связыванию с использованием RPB-ELISA. Можно оценить, что когда значение люминесценции, наблюдаемое в RPB-ELISA, составляет от 1,0 до 2,0 × 107, значение Kd составляет от 10-6 до 10-7 M; когда величина люминесценции, наблюдаемая в RPB-ELISA, составляет от 2,0 до 4,0 × 107, значение Kd составляет от 10-7 до 10-8 М; и когда значение люминесценции, наблюдаемое в RPB-ELISA, превышает 4,0 × 107, значение Kd составляет ~10-8 или ниже.
[Фиг. 11] Вероятность успешного конструирования. Белки PPR для 72 видов последовательностей-мишеней получают с использованием мотива v2, и вероятность успешного конструирования рассчитывают с помощью RPB-ELISA. Из 72 видов белков PPR 63 вида (88%), по оценкам, имеют значение Kd10-8 M или ниже (значение RPB-ELISA выше 1×107). Кроме того, 54 (75%) из них имеют значение специфичности (S/N) выше 10, это значение предназначено для оценки специфичности и рассчитывается путем деления сигнала связывания мишени на сигнал связывания, не являющегося мишенью. Эти результаты показывают, что через получение белка PPR с использованием мотива v2 можно эффективно получить специфичные для последовательности РНК-связывающие белки.
[Фиг. 12] Оценка активности связывания мишени в зависимости от количества мотивов PPR. A: Результаты для соответствующих последовательностей-мишеней. B: Средние значения для 18, 15 и 12 мотивов. Было определено, что большее количество мотивов обеспечивает более высокую силу связывания, и когда сравниваются те из 18 мотивов и 15 мотивов, белок с высокой силой связывания может быть стабильно получен с 18 мотивами.
[Фиг. 13] Пример искусственного контроля сплайсинга с белками PPR. A: Экспериментальная схема. Последовательности из 18 нуклеотидов выбирают из областей интрона 1, экзона 2 и интрона 2, и был проведен эксперимент для определения того, можно ли изменить соотношение количеств вариантов сплайсинга репортера RG-6 в зависимости от связывания белков PPR с последовательностями. B: изображения флуоресценции GFP и RFP клеток, полученные после культивирования с ДНК плазмидой экспрессии PPR и ДНК репортерной плазмидой RG-6. C: Соотношение вариантов сплайсинга. Полную РНК экстрагируют из клеток после получения изображений флуоресценции и электрофореза продуктов амплификации РВ-ПЦР. Измеряют интенсивности полосы около 114 п.н., которую рассматривают как полосу продукта ПЦР (а), полученную с проскоком экзонов, и полосу около 142 п.н., которую рассматривают как полосу продукта ПЦР (b), полученную без проскоков. Коэффициент сплайсинга рассчитывают как a/(a+b). Было определено, что коэффициент сплайсинга значительно изменяется при введении PPR. Было подтверждено, что проскок экзона можно изменить с помощью белков PPR, и, кроме того, было определено, что сплайсинг можно более эффективно изменить с помощью мотива v2.
[Фиг. 14] Влияние первого мотива PPR от N-конца на агрегацию. Каждый белок PPR получают в системе экспрессии Escherichia coli (E. coli ), очищают и разделяют гель-фильтрационной хроматографией. Меньший объем фракции элюирования (объем элюирования) указывает на больший размер молекулы. Те, которые используют v2, элюируют во фракциях элюирования от 8 до 10 мл, в то время как пик элюирования наблюдают для фракций элюирования от 12 до 14 мл с v3.2. Это предполагает возможность агрегации белков из-за большего размера белка, полученного с помощью v2, и было определено, что агрегация была улучшена с v3.2.
Способы осуществления изобретения
[Мотив PPR и белок PPR]
(Определение)
Мотив PPR, упоминаемый в настоящем изобретении, означает полипептид, состоящий из 30-38 аминокислот и имеющий аминокислотную последовательность со значением E, не превышающим предварительно определенное значение (желательно E-03), полученное для PF01535 в Pfam или PS51375 в Prosite, по данным анализа аминокислотной последовательности с помощью программы поиска белковых доменов в Интернете, если не указано иное. Номера положений аминокислот, составляющих мотив PPR, определенный в настоящем изобретении, по существу синонимичны позициям PF01535, и они соответствуют тем, которые получены вычитанием 2 из номеров аминокислотных положений PS51375 (например, положение 1, обозначенное в настоящем изобретении, соответствует положению 3 PS51375). Кроме того, термин «ii» (-2) аминокислота означает вторую аминокислоту от конца (сторона С-конца) аминокислот, составляющих мотив PPR, или вторую аминокислоту по направлению к стороне N-конца от первой аминокислоты следующего мотива PPR, т.е. вторую аминокислоту. Если следующий мотив PPR точно не идентифицирован, аминокислота 2 аминокислот перед первой аминокислотой следующей спиральной структуры является аминокислотой «ii». Для Pfam можно дать ссылку на http://pfam.sanger.ac.uk/, и для Prosite можно дать ссылку на http://www.expasy.org/prosite/.
Хотя консервативность консервативной аминокислотной последовательности мотива PPR низка на аминокислотном уровне, две из α-спиралей в качестве вторичной структуры являются высоко консервативными. Хотя типовой мотив PPR состоит из 35 аминокислот, его длина варьируется от 30 до 38 аминокислот.
Более конкретно, мотив PPR, упоминаемый в настоящем изобретении, состоит из полипептида длиной от 30 до 38 аминокислот, представленного формулой 1.
[Формула 1]
(Спираль А)-Х-(Спираль В)-L (формула 1)
В формуле:
Спираль A является группой длиной 12 аминокислот, способной образовывать структуру α-спирали, и представлена формулой 2;
[Формула 2]
А1-А2-А3-А4-А5-А6-А7-А8-А9-А10-А11-А12 (формула 2)
где в формуле 2, A1-A12 независимо являются аминокислотой;
X не существует или является группой длиной от 1 до 9 аминокислот;
Спираль B является группой длиной от 11 до 13 аминокислот, способной образовывать структуру α-спирали; и
L является группой длиной от 2 до 7 аминокислот, представленной формулой 3;
[Формула 3]
Lvii-Lvi-Lv-Liv-Liii-Lii-Li (формула 3)
где в формуле 3 аминокислоты пронумерованы «i» (-1), «ii» (-2) и так далее со стороны С-конца,
при условии, что Liii-Lvii могут не существовать.
Термин «белок PPR», используемый в настоящем изобретении, относится к белку PPR, содержащему один или несколько, предпочтительно два или несколько вышеупомянутых мотивов PPR, если не указано иное. Термин «белок», используемый в этом описании, относится к любому веществу, состоящему из полипептида (цепи, состоящей из множества аминокислот, связанных пептидными связями), если специально не указано иное, и включает вещества, состоящие из полипептида со сравнительно низкой молекулярной массой. Термин «аминокислота», используемый в настоящем изобретении, относится к обычной аминокислотной молекуле, а также относится к аминокислотному остатку, составляющему пептидную цепь. Который из них упоминается, должно быть ясно специалистам в данной области техники из контекста.
В настоящем изобретении термин специфичность/специфичный, используемый для свойства связывания мотива PPR с основанием в нуклеиновой кислоте-мишени, означает, что активность связывания с любым одним из четырех оснований выше, чем активности связывания с другими основаниями, если особо не указано иное.
В настоящем изобретении, термин нуклеиновая кислота относится к РНК или ДНК. Хотя белок PPR может обладать специфичностью к основаниям в РНК или ДНК, он не связывается с мономерами нуклеиновых кислот.
В мотиве PPR комбинация трех из 1, 4 и ii аминокислот важна для специфического связывания с основанием, и с каким основанием связывается мотив, можно определить в соответствии с этой комбинацией (вышеупомянутые патентные документы 1 и 2).
В частности, что касается РНК-связывающих мотивов PPR, соотношение между комбинациями трех из 1, 4 и ii аминокислот и оснований, с которыми они могут связываться, выглядит следующим образом (см. вышеупомянутый Патентный документ 1).
(3-1) Когда комбинация трех аминокислот A1, A4 и Lii состоит из валина, аспарагина и аспарагиновой кислоты в этом порядке, мотив PPR обладает такой селективной способностью связывать основания РНК, что мотив сильно связывается с U, менее сильно с C и еще менее с A или G.
(3-2) Когда комбинация трех аминокислот A1, A4 и Lii состоит из валина, треонина и аспарагина в этом порядке, мотив PPR обладает такой селективной способностью связывать основания РНК, что мотив сильно связывается с A, менее сильно с G и еще менее сильно с C, но не связывается с U.
(3-3) Когда комбинация трех аминокислот A1, A4 и Lii состоит из валина, аспарагина и аспарагина в этом порядке, мотив PPR обладает такой селективной способностью связывать основания РНК, что мотив сильно связывается с C и менее сильно с A или U, но не связывается с G.
(3-4) Когда комбинация трех аминокислот A1, A4 и Lii состоит из глутаминовой кислоты, глицина и аспарагиновой кислоты в этом порядке, мотив PPR обладает такой селективной способностью связывать основания РНК, что мотив сильно связывается с G, но не связывается с A, U и C.
(3-5) Когда комбинация трех аминокислот A1, A4 и Lii состоит из изолейцина, аспарагина и аспарагина в этом порядке, мотив PPR обладает такой селективной способностью связывать основания РНК, что мотив сильно связывается с C, менее сильно с U и еще менее сильно с A, но не связывается с G.
(3-6) Когда комбинация трех аминокислот A1, A4 и Lii состоит из валина, треонина и аспарагиновой кислоты в этом порядке, мотив PPR обладает такой селективной способностью связывать основания РНК, что мотив сильно связывается с G и менее сильно с U, но не связывается с A и C.
(3-7) Когда комбинация трех аминокислот A1, A4 и Lii состоит из лизина, треонина и аспарагиновой кислоты в этом порядке, мотив PPR обладает такой селективной способностью связывать основания РНК, что мотив сильно связывается с G и менее сильно с A, но не связывается с U и C.
(3-8) Когда комбинация трех аминокислот A1, A4 и Lii состоит из фенилаланина, серина и аспарагина в этом порядке, мотив PPR обладает такой селективной способностью связывать основания РНК, что мотив сильно связывается с A, менее сильно с C и еще менее сильно с G и U.
(3-9) Когда комбинация трех аминокислот A1, A4 и Lii состоит из валина, аспарагина и серина в этом порядке, мотив PPR обладает такой селективной способностью связывать основания РНК, что мотив сильно связывается с C и менее сильно с U, но не связывается с A и G.
(3-10) Когда комбинация трех аминокислот A1, A4 и Lii состоит из фенилаланина, треонина и аспарагина в этом порядке, мотив PPR обладает такой селективной способностью связывать основания РНК, что мотив сильно связывается с A, но не связывается с G, U и C.
(3-11) Когда комбинация трех аминокислот A1, A4 и Lii состоит из изолейцина, аспарагина и аспарагиновой кислоты в этом порядке, мотив PPR обладает такой селективной способностью связывать основания РНК, что мотив сильно связывается с U и менее сильно с A, но не связывается с G и C.
(3-12) Когда комбинация трех аминокислот A1, A4 и Lii состоит из треонина, треонина и аспарагина в этом порядке, мотив PPR обладает такой селективной способностью связывать основания РНК, что мотив сильно связывается с A, но не связывается с G, U и C.
(3-13) Когда комбинация трех аминокислот A1, A4 и Lii состоит из изолейцина, метионина и аспарагиновой кислоты в этом порядке, мотив PPR обладает такой селективной способностью связывать основания РНК, что мотив сильно связывается с U и менее сильно с C, но не связывается с A и G.
(3-14) Когда комбинация трех аминокислот A1, A4 и Lii состоит из фенилаланина, пролина и аспарагиновой кислоты в этом порядке, мотив PPR обладает такой селективной способностью связывать основания РНК, что мотив сильно связывается с U и менее сильно с C, но не связывается с A и G.
(3-15) Когда комбинация трех аминокислот A1, A4 и Lii состоит из тирозина, пролина и аспарагиновой кислоты в этом порядке, мотив PPR обладает такой селективной способностью связывать основания РНК, что мотив сильно связывается с U, но не связывается с A, G и C.
(3-16) Когда комбинация трех аминокислот A1, A4 и Lii состоит из лейцина, треонина и аспарагиновой кислоты в этом порядке, мотив PPR обладает такой селективной способностью связывать основания РНК, что мотив сильно связывается с G, но не связывается с A, U и C.
В частности, что касается ДНК-связывающих мотивов PPR, соотношение между комбинациями трех из 1, 4 и ii аминокислот и оснований, с которыми они могут связываться, выглядит следующим образом (см. вышеупомянутый Патентный документ 2).
(2-1) Когда комбинация трех аминокислот A1, A4 и Lii состоит из произвольной аминокислоты, глицина и аспарагиновой кислоты в этом порядке, мотив PPR селективно связывается с G.
(2-2) Когда комбинация трех аминокислот A1, A4 и Lii состоит из глутаминовой кислоты, глицина и аспарагиновой кислоты в этом порядке, мотив PPR селективно связывается с G.
(2-3) Когда комбинация трех аминокислот A1, A4 и Lii состоит из произвольной аминокислоты, глицина и аспарагина в этом порядке, мотив PPR селективно связывается с A.
(2-4) Когда комбинация трех аминокислот A1, A4 и Lii состоит из глутаминовой кислоты, глицина и аспарагина в этом порядке, мотив PPR селективно связывается с A.
(2-5) Когда комбинация трех аминокислот A1, A4 и Lii состоит из произвольной аминокислоты, глицина и серина в этом порядке, мотив PPR селективно связывается с A и менее селективно с.
(2-6) Когда комбинация трех аминокислот A1, A4 и Lii состоит из произвольной аминокислоты, изолейцина и произвольной аминокислоты в этом порядке, мотив PPR селективно связывается с T и C .
(2-7) Когда комбинация трех аминокислот A1, A4 и Lii состоит из произвольной аминокислоты, изолейцина и аспарагина в этом порядке, мотив PPR селективно связывается с T и менее селективно с С.
(2-8) Когда комбинация трех аминокислот A1, A4 и Lii состоит из произвольной аминокислоты, лейцина и произвольной аминокислоты в этом порядке, мотив PPR селективно связывается с T и C .
(2-9) Когда комбинация трех аминокислот A1, A4 и Lii состоит из произвольной аминокислоты, лейцина и аспарагиновой кислоты в этом порядке, мотив PPR селективно связывается с C.
(2-10) Когда комбинация трех аминокислот A1, A4 и Lii состоит из произвольной аминокислоты, лейцина и лизина в этом порядке, мотив PPR селективно связывается с T.
(2-11) Когда комбинация трех аминокислот A1, A4 и Lii состоит из произвольной аминокислоты, метионина и произвольной аминокислоты в этом порядке, мотив PPR селективно связывается с T.
(2-12) Когда комбинация трех аминокислот A1, A4 и Lii состоит из произвольной аминокислоты, метионина и аспарагиновой кислоты в этом порядке, мотив PPR селективно связывается с T.
(2-13) Когда комбинация трех аминокислот A1, A4 и Lii состоит из изолейцина, метионина и аспарагиновой кислоты в этом порядке, мотив PPR селективно связывается с T и менее селективно с C.
(2-14) Когда комбинация трех аминокислот A1, A4 и Lii состоит из произвольной аминокислоты, аспарагина и произвольной аминокислоты в этом порядке, мотив PPR селективно связывается с C и T.
(2-15) Когда комбинация трех аминокислот A1, A4 и Lii состоит из произвольной аминокислоты, аспарагина и аспарагиновой кислоты в этом порядке, мотив PPR селективно связывается с T.
(2-16) Когда комбинация трех аминокислот A1, A4 и Lii состоит из фенилаланина, аспарагина и аспарагиновой кислоты в этом порядке, мотив PPR селективно связывается с T.
(2-17) Когда комбинация трех аминокислот A1, A4 и Lii состоит из глицина, аспарагина и аспарагиновой кислоты в этом порядке, мотив PPR селективно связывается с T.
(2-18) Когда комбинация трех аминокислот A1, A4 и Lii состоит из изолейцина, аспарагина и аспарагиновой кислоты в этом порядке, мотив PPR селективно связывается с T.
(2-19) Когда комбинация трех аминокислот A1, A4 и Lii состоит из треонина, аспарагина и аспарагиновой кислоты в этом порядке, мотив PPR селективно связывается с T.
(2-20) Когда комбинация трех аминокислот A1, A4 и Lii состоит из валина, аспарагина и аспарагиновой кислоты в этом порядке, мотив PPR селективно связывается с T и менее селективно с C.
(2-21) Когда комбинация трех аминокислот A1, A4 и Lii состоит из тирозина, аспарагина и аспарагиновой кислоты в этом порядке, мотив PPR селективно связывается с T и менее селективно с C.
(2-22) Когда комбинация трех аминокислот A1, A4 и Lii состоит из произвольной аминокислоты, аспарагина и аспарагина в этом порядке, мотив PPR селективно связывается с C.
(2-23) Когда комбинация трех аминокислот A1, A4 и Lii состоит из изолейцина, аспарагина и аспарагина в указанном порядке, мотив PPR селективно связывается с C.
(2-24) Когда комбинация трех аминокислот A1, A4 и Lii состоит из серина, аспарагина и аспарагина в этом порядке, мотив PPR селективно связывается с C.
(2-25) Когда комбинация трех аминокислот A1, A4 и Lii состоит из валина, аспарагина и аспарагина в этом порядке, мотив PPR селективно связывается с C.
(2-26) Когда комбинация трех аминокислот A1, A4 и Lii состоит из произвольной аминокислоты, аспарагина и серина в этом порядке, мотив PPR селективно связывается с C.
(2-27) Когда комбинация трех аминокислот A1, A4 и Lii состоит из валина, аспарагина и серина в этом порядке, мотив PPR селективно связывается с C.
(2-28) Когда комбинация трех аминокислот A1, A4 и Lii состоит из произвольной аминокислоты, аспарагина и треонина в этом порядке, мотив PPR селективно связывается с C.
(2-29) Когда комбинация трех аминокислот A1, A4 и Lii состоит из валина, аспарагина и треонина в этом порядке, мотив PPR селективно связывается с C.
(2-30) Когда комбинация трех аминокислот A1, A4 и Lii состоит из произвольной аминокислоты, аспарагина и триптофана в этом порядке, мотив PPR селективно связывается с C и менее селективно с Т.
(2-31) Когда комбинация трех аминокислот A1, A4 и Lii состоит из изолейцина, аспарагина и триптофана в этом порядке, мотив PPR селективно связывается с T и менее селективно с C.
(2-32) Когда комбинация трех аминокислот A1, A4 и Lii состоит из произвольной аминокислоты, пролина и произвольной аминокислоты в этом порядке, мотив PPR селективно связывается с T.
(2-33) Когда комбинация трех аминокислот A1, A4 и Lii состоит из произвольной аминокислоты, пролина и аспарагиновой кислоты в этом порядке, мотив PPR селективно связывается с T.
(2-34) Когда комбинация трех аминокислот A1, A4 и Lii состоит из фенилаланина, пролина и аспарагиновой кислоты в этом порядке, мотив PPR селективно связывается с T.
(2-35) Когда комбинация трех аминокислот A1, A4 и Lii состоит из тирозина, пролина и аспарагиновой кислоты в этом порядке, мотив PPR селективно связывается с T.
(2-36) Когда комбинация трех аминокислот A1, A4 и Lii состоит из произвольной аминокислоты, серина и произвольной аминокислоты в этом порядке, мотив PPR селективно связывается с A и G.
(2-37) Когда комбинация трех аминокислот A1, A4 и Lii состоит из произвольной аминокислоты, серина и аспарагина в этом порядке, мотив PPR селективно связывается с A.
(2-38) Когда комбинация трех аминокислот A1, A4 и Lii состоит из фенилаланина, серина и аспарагина в этом порядке, мотив PPR селективно связывается с A.
(2-39) Когда комбинация трех аминокислот A1, A4 и Lii состоит из валина, серина и аспарагина в этом порядке, мотив PPR селективно связывается с A.
(2-40) Когда комбинация трех аминокислот A1, A4 и Lii состоит из произвольной аминокислоты, треонина и произвольной аминокислоты в этом порядке, мотив PPR селективно связывается с A и G.
(2-41) Когда комбинация трех аминокислот A1, A4 и Lii состоит из произвольной аминокислоты, треонина и аспарагиновой кислоты в этом порядке, мотив PPR селективно связывается с G.
(2-42) Когда комбинация трех аминокислот A1, A4 и Lii состоит из валина, треонина и аспарагиновой кислоты в этом порядке, мотив PPR селективно связывается с G.
(2-43) Когда комбинация трех аминокислот A1, A4 и Lii состоит из произвольной аминокислоты, треонина и аспарагина в этом порядке, мотив PPR селективно связывается с A.
(2-44) Когда комбинация трех аминокислот A1, A4 и Lii состоит из фенилаланина, треонина и аспарагина в указанном порядке, мотив PPR селективно связывается с A.
(2-45) Когда комбинация трех аминокислот A1, A4 и Lii состоит из изолейцина, треонина и аспарагина в этом порядке, мотив PPR селективно связывается с A.
(2-46) Когда комбинация трех аминокислот A1, A4 и Lii состоит из валина, треонина и аспарагина в этом порядке, мотив PPR селективно связывается с A.
(2-47) Когда комбинация трех аминокислот A1, A4 и Lii состоит из произвольной аминокислоты, валина и произвольной аминокислоты в этом порядке, мотив PPR связывается с A, C, и T, но не связывается с G.
(2-48) Когда комбинация трех аминокислот A1, A4 и Lii состоит из изолейцина, валина и аспарагиновой кислоты в этом порядке, мотив PPR селективно связывается с C и менее селективно с A.
(2-49) Когда комбинация трех аминокислот A1, A4 и Lii состоит из произвольной аминокислоты, валина и глицина в этом порядке, мотив PPR селективно связывается с C.
(2-50) Когда комбинация трех аминокислот A1, A4 и Lii состоит из произвольной аминокислоты, валина и треонина в этом порядке, мотив PPR селективно связывается с T.
(Новые мотивы PPR)
Настоящее изобретение представляет новые мотивы PPR. Новые мотивы PPR, обладающие свойством связывания аденина, представляемые настоящим изобретением, являются (A-1), (A-2) и (A-3), указанными ниже:
(A-1) мотив PPR, состоящий из последовательности SEQ ID NO: 9, или мотив PPR, состоящий из последовательности SEQ ID NO: 9, имеющий замену, выбранную из группы, состоящей из аминокислотной замены в положении 10 тирозином, аминокислотной замены в положении 15 лизином, аминокислотной замены в положении 16 лейцином, аминокислотной замены в положении 17 глутаминовой кислотой, аминокислотной замены в положении 18 аспарагиновой кислотой и аминокислотной замены в положении 28 глутаминовой кислотой;
(A-2) мотив PPR, состоящий из последовательности SEQ ID NO: 9, имеющей замену, делецию или добавление от 1 до 20 аминокислот, кроме аминокислот в положениях 1, 2, 3, 4, 6, 7, 9, 11, 12, 14, 19, 26, 30, 33 и 34, и обладающий свойством связывания аденина; и
(A-3) мотив PPR, имеющий идентичность последовательности, по меньшей мере, 42% с последовательностью SEQ ID NO: 9, при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 7, 9, 11, 12, 14, 19, 26, 30, 33 и 34 идентичны, и обладающий свойством связывания аденина;
Замена в мотиве (A-1) может состоять из одной, двух или более или всех замен, упомянутых выше.
В мотиве (A-2), 1-20 аминокислотами, отличными от аминокислот в положениях 1, 2, 3, 4, 6, 7, 9, 11, 12, 14, 19, 26, 30, 33, и 34, которыми являются аминокислоты в последовательности SEQ ID NO: 9, которые могут быть заменены, или подобными, являются:
предпочтительно, 1-11 аминокислот, отличных от аминокислот в положениях 1, 2, 3, 4, 6, 7, 9, 11, 12, 14, 19, 26, 30, 33 и 34, и отличных от аминокислот в положениях 5, 8, 13, 21, 22, 23, 25, 29 и 35,
более предпочтительно, 1-7 аминокислот, отличных от аминокислот в положениях 1, 2, 3, 4, 6, 7, 9, 11, 12, 14, 19, 26, 30, 33 и 34, отличных от аминокислот в положениях 5, 8, 13, 21, 22, 23, 25, 29 и 35 и отличных от аминокислот в положениях 20, 24, 31 и 32,
более предпочтительно, любая из аминокислот в положениях 10, 15, 16, 17, 18 и 28.
Характеристика мотива (A-3), имеющего идентичность последовательности, по меньшей мере, 42% с последовательностью SEQ ID NO: 9, при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 7, 9, 11, 12, 14, 19, 26, 30, 33 и 34 идентичны:
предпочтительно, иметь идентичность последовательности, по меньшей мере, 71% с последовательностью SEQ ID NO: 9 при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 7, 9, 11, 12, 14, 19, 26, 30, 33 и 34, и аминокислоты в положениях 5, 8, 13, 21, 22, 23, 25, 29 и 35 идентичны,
более предпочтительно, иметь идентичность последовательности, по меньшей мере, 80% с последовательностью SEQ ID NO: 9 при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 7, 9, 11, 12, 14, 19, 26, 30, 33 и 34, аминокислоты в положениях 5, 8, 13, 21, 22, 23, 25, 29 и 35, и аминокислоты в положениях 20, 24, 31 и 32 идентичны,
еще более предпочтительно, иметь идентичность последовательности, по меньшей мере, 82% с последовательностью SEQ ID NO: 9 при условии, что не идентичной аминокислотой является любая из аминокислот в положениях 10, 15, 16, 17, 18 и 28.
Новые мотивы PPR и обладающие свойством связывания цитозина, представленные настоящим изобретением, являются (C-1), (C-2) и (C-3), указанными ниже:
(C-1) мотив PPR, состоящий из последовательности SEQ ID NO: 10, или мотив PPR, состоящий из последовательности SEQ ID NO: 10, имеющей замену аминокислоты, выбранной из группы, состоящей из аминокислотной замены в положении 2 серином, аминокислотной замены в положении 5 изолейцином, аминокислотной замены в положении 7 лейцином, аминокислотной замены в положении 8 лизином, аминокислотной замены в положении 10 фенилаланином или тирозином, аминокислотной замены в положении 15 аргинином, аминокислотной замены в положении 22 валином, аминокислотной замены в положении 24 аргинином, аминокислотной замены в положении 27 лейцином и аминокислотной замены в положении 29 аргинином;
(C-2) мотив PPR, состоящий из последовательности SEQ ID NO: 10, имеющей замену, делецию или добавление 1-25 аминокислот, кроме аминокислот в положениях 1, 3, 4, 14, 18, 19. , 26, 30, 33 и 34, и обладающий свойством связывания цитозина; и
(C-3) мотив PPR, имеющий идентичность последовательности, по меньшей мере, 25% с последовательностью SEQ ID NO: 10, при условии, что аминокислоты в положениях 1, 3, 4, 14, 18, 19, 26, 30, 33 и 34 идентичны, и обладающий свойством связывания цитозина;
Замена в мотиве (C-1) может состоять из одной, двух или более или всех упомянутых замен.
В мотиве (C-2), 1-25 аминокислотами, отличными от аминокислот в положениях 1, 3, 4, 14, 18, 19, 26, 30, 33 и 34, которые являются аминокислотами, которые могут быть заменены, или подобными, в последовательности SEQ ID NO: 10 являются
предпочтительно, 1-14 аминокислот, отличных от аминокислот в положениях 1, 3, 4, 14, 18, 19, 26, 30, 33 и 34, и отличных от аминокислот в положениях 6, 9, 11, 12, 17, 20, 21, 23, 25, 28 и 35,
более предпочтительно, 1-10 аминокислот отличных от аминокислот в положениях 1, 3, 4, 14, 18, 19, 26, 30, 33 и 34, отличных от аминокислот в положениях 6, 9, 11, 12, 17, 20, 21, 23, 25, 28 и 35, и отличных от аминокислот в положениях 13, 16, 31 и 32,
еще более предпочтительно, любая из аминокислот в положениях 2, 5, 7, 8, 10, 15, 22, 24, 27 и 29.
Характеристика мотива (C-3), имеющего идентичность последовательности, по меньшей мере, на 25% с последовательностью SEQ ID NO: 10, при условии, что аминокислоты в положениях 1, 3, 4, 14, 18, 19, 26 , 30, 33 и 34 идентичны:
предпочтительно, иметь идентичность последовательности, по меньшей мере, 60% с последовательностью SEQ ID NO: 10, при условии, что аминокислоты в положениях 1, 3, 4, 14, 18, 19, 26, 30, 33 и 34 и аминокислоты в положениях 6, 9, 11, 12, 17, 20, 21, 23, 25, 28 и 35 идентичны,
более предпочтительно, иметь идентичность последовательности, по меньшей мере, 71% с последовательностью SEQ ID NO: 10 при условии, что аминокислоты в положениях 1, 3, 4, 14, 18, 19, 26, 30, 33 и 34 , аминокислоты в положениях 6, 9, 11, 12, 17, 20, 21, 23, 25, 28 и 35 и аминокислоты в положениях 13, 16, 31 и 32 идентичны,
еще более предпочтительно, иметь идентичность последовательности, по меньшей мере, 71% с последовательностью SEQ ID NO: 10, при условии, что аминокислотой, которая не идентична, является любая из аминокислот в положениях 2, 5, 7, 8, 10, 15, 22, 24, 27 и 29.
Новые мотивы PPR, обладающие свойством связывания гуанина, представляемые настоящим изобретением, являются (G-1), (G-2) и (G-3), указанными ниже:
(G-1) мотив PPR, состоящий из последовательности SEQ ID NO: 11, или мотив PPR, состоящий из последовательности SEQ ID NO: 11, имеющий замену, выбранную из группы, состоящей из аминокислотной замены в положении 10 фенилаланином, аминокислотной замены в положении 15 аспарагиновой кислотой, аминокислотной замены в положении 27 валином, аминокислотной замены в положении 28 серином и аминокислотной замены в положении 35 изолейцином;
(G-2) мотив PPR, состоящий из последовательности SEQ ID NO: 11, имеющей замену, делецию или добавление от 1 до 21 аминокислот, кроме аминокислот в положениях 1, 2, 3, 4, 6, 7, 9, 14, 18, 19, 26, 30, 33 и 34, и обладающий свойством связывания гуанина; и
(G-3) мотив PPR, имеющий идентичность последовательности, по меньшей мере, 40% с последовательностью SEQ ID NO: 11, при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 7, 9, 14, 18, 19, 26, 30, 33 и 34 идентичны и обладают свойством связывания гуанина.
Замена в мотиве (G-1) может состоять из одной, двух или более или всех замен, упомянутых выше.
В мотиве (G-2), 1-21 аминокислотами, отличными от аминокислот в положениях 1, 2, 3, 4, 6, 7, 9, 14, 18, 19, 26, 30, 33 и 34, которыми являются аминокислоты, которые могут быть заменены, или подобными, в последовательности SEQ ID NO: 11, являются:
предпочтительно, 1-12 аминокислот, отличных от аминокислот в положениях 1, 2, 3, 4, 6, 7, 9, 14, 18, 19, 26, 30, 33 и 34, и отличных от аминокислот в положениях позиции 5, 11, 12, 17, 20, 21, 22, 23 и 25,
более предпочтительно, 1-5 аминокислот, отличных от аминокислот в положениях 1, 2, 3, 4, 6, 7, 9, 14, 18, 19, 26, 30, 33 и 34, отличных от аминокислот в положениях положения 5, 11, 12, 17, 20, 21, 22, 23 и 25, и отличных от аминокислот в положениях 8, 13, 16, 24, 29, 31 и 32,
еще более предпочтительно, любая из аминокислот в положениях 10, 15, 27, 28 и 35.
Характеристика мотива (G-3), имеющего идентичность последовательности, по меньшей мере, 40% с последовательностью SEQ ID NO: 11 при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 7, 9 , 14, 18, 19, 26, 30, 33 и 34 идентичны:
предпочтительно, иметь идентичность последовательности, по меньшей мере, 65% с последовательностью SEQ ID NO: 11, при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 7, 9, 14, 18, 19, 26 , 30, 33 и 34, и аминокислоты в положениях 5, 11, 12, 17, 20, 21, 22, 23 и 25 идентичны;
более предпочтительно, иметь идентичность последовательности, по меньшей мере, 85% с последовательностью SEQ ID NO: 11, при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 7, 9, 14, 18, 19, 26, 30, 33 и 34, аминокислоты в положениях 5, 11, 12, 17, 20, 21, 22, 23 и 25, и аминокислоты в положениях 8, 13, 16, 24, 29, 31 , и 32 идентичны,
еще более предпочтительно, иметь идентичность последовательности, по меньшей мере, 85% с последовательностью SEQ ID NO: 11, при условии, что не идентичной аминокислотой является любая из аминокислот в положениях 10, 15, 27, 28 и 35.
Новые мотивы PPR, обладающие свойством связывания урацила, представляемые настоящим изобретением, являются мотивы (U-1), (U-2) и (U-3), указанными ниже:
(U-1) мотив PPR, состоящий из последовательности SEQ ID NO: 12, или мотив PPR, состоящий из последовательности SEQ ID NO: 12, имеющий замену, выбранную из группы, состоящей из аминокислотной замены в положении 10 фенилаланином, аминокислотной замены в положении 13 серином, аминокислотной замены в положении 15 лизином, аминокислотной замены в положении 17 глутаминовой кислотой, аминокислотной замены в положении 20 лейцином, аминокислотные замены в положении 21 лизином, аминокислотной замены в положении 23 фенилаланином, аминокислотной замены в положении 24 аспарагиновой кислотой, аминокислотной замены в положении 27 лизином, аминокислотной замены в положении 28 лизином, аминокислотной замены в положении 29 аргинином и аминокислотной замены в положении 31 лейцином;
(U-2) мотив PPR, состоящий из последовательности SEQ ID NO: 12, имеющей замену, делецию или добавление от 1 до 22 аминокислот, кроме аминокислот в положениях 1, 2, 3, 4, 6, 11, 12, 14, 19, 26, 30, 33 и 34, и обладающий свойством связывания урацила; и
(U-3) мотив PPR, имеющий идентичность последовательности, по меньшей мере, 37% с последовательностью SEQ ID NO: 12, при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 11, 12, 14, 19, 26, 30, 33 и 34 идентичны, и обладающий свойством связывания урацила.
Замена в мотиве (U-1) может состоять из одной, двух или более или всех замен, упомянутых выше.
В мотиве (U-2), 1-22 аминокислотами, отличными от аминокислот в положениях 1, 2, 3, 4, 6, 11, 12, 14, 19, 26, 30, 33 и 34, которые являются аминокислотами, которые могут быть заменены, и подобными в последовательности SEQ ID NO: 12, являются
предпочтительно, 1-14 аминокислот, отличных от аминокислот в положениях 1, 2, 3, 4, 6, 11, 12, 14, 19, 26, 30, 33 и 34, и отличных от аминокислот в положениях 5, 7, 9, 16, 18, 22, 25 и 35,
более предпочтительно, 1-12 аминокислот, отличных от аминокислот в положениях 1, 2, 3, 4, 6, 11, 12, 14, 19, 26, 30, 33 и 34, отличных от аминокислот в положениях 5, 7, 9, 16, 18, 22, 25 и 35, и отличных от аминокислот в положениях 8 и 32,
еще более предпочтительно, любая из аминокислот в положениях 10, 13, 15, 17, 20, 21, 23, 24, 27, 28, 29 и 31.
Характеристика мотива (U-3), имеющего идентичность последовательности, по меньшей мере, 37% с последовательностью SEQ ID NO: 12, при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 11, 12 , 14, 19, 26, 30, 33 и 34 идентичны, включает:
предпочтительно, иметь идентичность последовательности, по меньшей мере, 60% с последовательностью SEQ ID NO: 12 при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 11, 12, 14, 19, 26, 30 , 33 и 34, и аминокислоты в положениях 5, 7, 9, 16, 18, 22, 25 и 35 идентичны;
более предпочтительно, иметь идентичность последовательности, по меньшей мере, 65% с последовательностью SEQ ID NO: 12 при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 11, 12, 14, 19, 26, 30, 33 и 34, аминокислоты в положениях 5, 7, 9, 16, 18, 22, 25 и 35 и аминокислоты в положениях 8 и 32 идентичны,
еще более предпочтительно, иметь идентичность последовательности, по меньшей мере, 65% с последовательностью SEQ ID NO: 12 при условии, что не идентичной аминокислотой является любая из аминокислот в положениях 10, 13, 15, 17, 20, 21, 23, 24, 27, 28, 29 и 31.
Мотивы PPR v2_A (SEQ ID NO: 9), v2_C (SEQ ID NO: 10), v2_G (SEQ ID NO: 11) и v2_U (SEQ ID NO: 12), которые были созданы авторами настоящего изобретения, впервые описаны в этой заявке и не существуют в природе. Что касается их гомологов (варианты осуществления, упомянутые выше как (A-1), (A-2), (A-3), (C-1), (C-2), (C-3), (G-1), (G-2), (G-3), (U-1), (U-2) и (U-3) и их предпочтительные варианты осуществления, которые содержат последовательность, отличную от последовательностей SEQ ID NO: 9-12), считается, что комбинации, по меньшей мере, любых двух или нескольких, например, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13 или 14, гомологов не существуют в природе (независимо от того, описаны ли отдельные гомологи впервые в этой заявке и существуют ли они в природе). В настоящем изобретении число, обозначенное термином «любое», может быть произвольным числом.
(Объяснение последовательностей новых мотивов PPR)
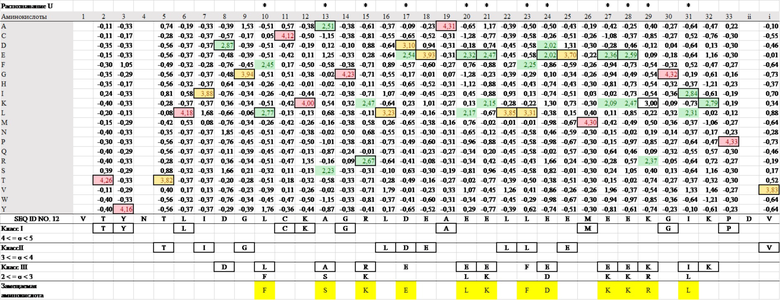
На фиг. 1-4 суммированы типы и встречающееся количество аминокислот в каждом положении в последовательностях мотива PPR Arabidopsis thaliana, для которых были собраны мотивы PPR, в которых комбинация аминокислот, расположенных в положениях 1, 4 и ii, является VTN в качестве аденин-распознающих мотивов PPR, является VSN в качестве цитозин-распознающих мотивов PPR, является VTD в качестве гуанин-распознающих мотивов PPR, VND, в качестве урацил-распознающих мотивов PPR. Аминокислоты во всех положениях последовательностей новых мотивов PPR v2_A (SEQ ID NO: 9), v2_C (SEQ ID NO: 10), v2_G (SEQ ID NO: 11) и v2_U (SEQ ID NO: 12) являются наиболее часто встречающимися. На фиг. 6A, наряду с этими новыми последовательностями, также показаны v1_A (SEQ ID NO: 13), v1_C (SEQ ID NO: 14), v1_G (SEQ ID NO: 15) и v1_U (SEQ ID NO: 16), последовательности которых имеют мотив dPPR, в котором комбинация аминокислот в положениях 1, 4 и ii такая же, как у v2.
На фиг. 6А также показана аминокислотная последовательность мотива v3.1. v3.1 совпадает с v2, за исключением того, что мутация D15K вводится в аденин-распознающий мотив v2 (SEQ ID NO: 401), и, таким образом, другие их части идентичны. Использование v3.1 в белках PPR может дать такие, которые демонстрируют улучшенную связывающую способность по сравнению с v2.
Кроме того, в таблицах 1-4, представленных ниже, суммированы величины отклонений частоты встречаемости аминокислот в последовательностях SEQ ID NO: 9-12 от случайного появления (например, если собрано 100 мотивов PPR и предполагается, что аминокислоты случайным образом встречаются в определенном положении, что касается частоты встречаемости аминокислот, каждый из 20 типов аминокислот должен встречаться в этом положении 5 раз). Если частота встречаемости аминокислоты в определенном положении отличается от случайной и высока, считается, что аминокислота в этом положении эволюционно конвергирована и высоко родственна функции. Даже если аминокислота заменена другим типом аминокислоты, частота встречаемости которой отклоняется от случайной встречаемости и частота встречаемости которой высока, функция мотива PPR может сохраняться, пока аминокислота в высокой степени родственна функции.
[Таблица 1]
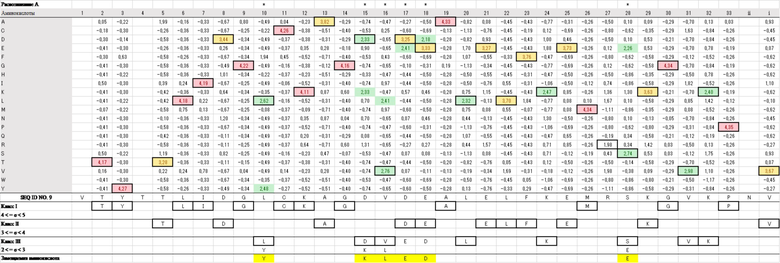
[Таблица 2]
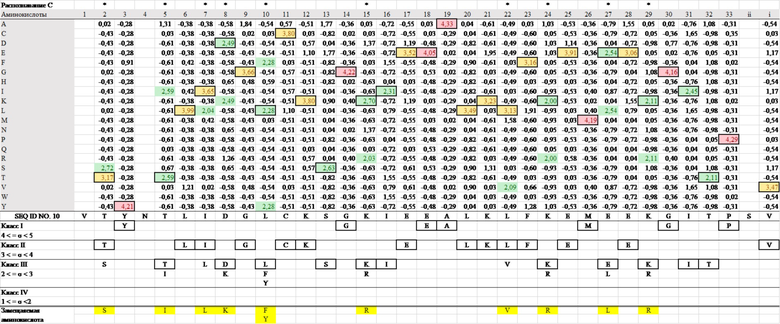
[Таблица 3]
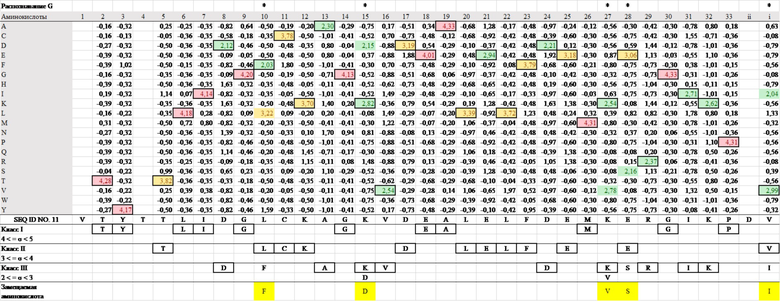
[Таблица 4]
(Новый белок PPR)
Настоящее изобретение представляет новые белки PPR, содержащие новый мотив PPR.
Новые белки PPR, представленные в настоящем изобретении, упомянуты ниже.
Белок, содержащий n мотивов PPR и способный связываться с РНК-мишенью, состоящей из последовательности из n оснований длиной, где:
мотивом PPR для аденина в нуклеотидной последовательности является мотив PPR (A-1), (A-2) или (A-3), упомянутый выше;
мотивом PPR для цитозина в нуклеотидной последовательности является мотив PPR (C-1), (C-2) или (c-3), упомянутый выше;
мотивом PPR для гуанина в нуклеотидной последовательности является мотив PPR (G-1), (G-2) или (G-3), упомянутый выше; и
мотивом PPR для урацила в нуклеотидной последовательности является мотив PPR (U-1), (U-2) или (U-3), упомянутый выше.
Что касается предпочтительных примеров мотивов PPR, содержащихся в белках PPR, описания, касающиеся мотивов PPR для (A-1), (A-2), (A-3), (C-1), (C-2), (C-3), (G-1), (G-2), (G-3), (U-1), (U-2) или (U-3), упомянутых выше, могут применяться как есть.
В белке PPR по настоящему изобретению n (представляющее целое число 1 или больше) особо не ограничивается, но может быть 10 или больше, предпочтительно, 12 или больше, более предпочтительно, 15 или больше, еще более предпочтительно, 18 или больше. Увеличенное количество мотивов позволяет получить белок PPR, демонстрирующий высокую силу связывания с большим количеством видов мишеней.
Хотя до сих пор сообщалось о получении искусственных белков PPR, содержащих от 7 до 14 мотивов, как показано в таблицах, упомянутых ниже, считалось, что конструирование гена для белка PPR, содержащего большее количество мотивов PPR, неизбежно должно приводить к большему количеству повторов в нуклеотидной последовательности гена, кодирующей такой белок. В общем, может быть трудно получить гены, содержащие повторяющиеся последовательности, потому что, например, повторяющиеся группы рекомбинируются в процессе клонирования (Trinh, T. et al., An Escherichia coli strain for the stable propagation of retroviral clones and direct repeat sequences, Focus, 16, 78-80 (1994)).
В таблице значения Kd являются самыми низкими среди значений, приведенных в литературе.
[Таблица 5]

При конструировании гена для белка PPR, имеющего 15 или более мотивов PPR, ген, в котором количество повторов снижено, может быть сконструирован путем соответствующего изменения нуклеотидных последовательностей, кодирующих аминокислоты, отличные от аминокислот в положениях 1, 4 и ii, ответственных за связывание (при использовании описанного ниже способа Golden Gate, 29 аминокислот с 5-й по 33-ю, отличных от общих обоих концевых областей) среди мотивов с использованием вырожденности кодонов. Величина изменения может быть соответствующим образом определена специалистами в данной области техники, и, например, 4,5% или более (более 4 положений в 87 основаниях), 15% или более или 30% или более (более 26 положений в 87 базах) оснований можно менять.
Например, примеры нуклеотидных последовательностей, кодирующих мотив, полученных с использованием вырожденности кодонов из существующих последовательностей, кодирующих мотивы от v1 до v4 (SEQ ID NO: 13-16), включают последовательности, показанные в таблице, представленной ниже.
[Таблица 6]
Термин «получение» можно перефразировать как «производство» или «изготовление». Кроме того, термин «конструирование» иногда используют для обозначения получения гена или подобного путем объединения частей, и «конструирование» также можно перефразировать как «производство» или «производство».
(Нуклеиновые кислоты, кодирующие мотив PPR и белок PPR)
Настоящее изобретение представляет нуклеиновые кислоты, кодирующие новые мотивы PPR, и новые белки PPR, содержащие эти мотивы. Существует несколько вариантов последовательностей нуклеиновых кислот, кодирующих новые мотивы PPR, из-за вырожденности кодонов.
Предпочтительные примеры нуклеотидных последовательностей, кодирующих аминокислотные последовательности новых мотивов PPR по настоящему изобретению, v2_A (SEQ ID NO: 9), v2_C (SEQ ID NO: 10), v2_G (SEQ ID NO: 11) и v2_U ( SEQ ID NO: 12) показаны в таблице, упомянутой ниже.
[Таблица 7-1]
Нуклеотидные последовательности, кодирующие аминокислотные последовательности мотивов PPR v1_A (SEQ ID NO: 13), v1_C (SEQ ID NO: 14), v1_G (SEQ ID NO: 15) и v1_U (SEQ ID NO: 16), которые соответствуют мотиву dPPR, имеющему такую же комбинацию аминокислот в положениях 1, 4 и ii, что и v2, показаны в таблице, упомянутой ниже.
[Таблица 7-2]
Нуклеотидная последовательность, кодирующая белок PPR, может состоять из любой комбинации последовательностей, упомянутых выше. Нуклеотидная последовательность, кодирующая аминокислоты белка, может быть составлена соответствующим объединением нуклеотидных последовательностей, кодирующих аминокислотные последовательности v2_A (SEQ ID NO: 9), v2_C (SEQ ID NO: 10), v2_G (SEQ ID NO: 11) и v2_U (SEQ ID NO: 12), и нуклеотидные последовательности, кодирующие аминокислотные последовательности v1_A (SEQ ID NO: 13), v1_C (SEQ ID NO: 14), v1_G (SEQ ID NO: 15) и v1_U ( SEQ ID NO: 16).
Предпочтительные примеры нуклеотидных последовательностей, кодирующих аминокислотные последовательности v3.1_A (SEQ ID NO: 401), 1st_A (SEQ ID NO: 402), 1st_C (SEQ ID NO: 403), 1st_G (SEQ ID NO: 404) и 1st_U (SEQ ID NO: 405) новых мотивов PPR по настоящему изобретению показаны в таблице, представленной ниже.
[Таблица 8]
(1st_A)
(1st_C)
(1st_G)
(1st_U)
Нуклеотидная последовательность, кодирующая белок PPR, может состоять из любой комбинации последовательностей, упомянутых выше. Можно выбрать любую последовательность, выбранную из v3.2_X, упомянутых выше, в качестве нуклеотидной последовательности, кодирующей первый мотив PPR от N-конца, затем для нуклеотидных последовательностей, кодирующих следующие мотивы PPR, v3.1_A, упомянутую выше, в качестве нуклеотидной последовательности, кодирующей мотив PPR для аденина, и нуклеотидные последовательности, выбранные из серии v2, упомянутой выше, в качестве нуклеотидных последовательностей, кодирующих мотивы PPR для цитозина, гуанина и урацила, и их можно подходящим образом комбинировать.
(Улучшение свойства агрегации)
Авторы настоящего изобретения обнаружили, что аминокислота в положении 6 мотива PPR очень часто является гидрофобной аминокислотой (особенно лейцином), а аминокислота в положении 9 чрезвычайно часто является не гидрофильной аминокислотой (особенно глицином) на основе аминокислотной информации существующих природных мотивов PPR. На основе структур белков PPR, для которых уже получены кристаллические структуры (не патентный документ 6: Coquille et al., 2014 Nat. Commun., PDB ID: 4PJQ, 4WN4, 4WSL, 4PJR, не патентный документ 7: Shen et al., 2015 Nat. Commun., PDB ID: 519D, 5I9F, 5I9G, 5I9H), было сделано предположение, что, поскольку эти 6 и 9 аминокислоты в первом мотиве (со стороны N-конца) подвергаются воздействию извне, белки проявляют свойство агрегации за счет этих подвергшихся воздействию гидрофобных аминокислот (фиг. 6A). С другой стороны, считается, что во втором и последующих мотивах, 6 и 9 аминокислоты скрыты внутри белка и образуют гидрофобное ядро, и, следовательно, если гидрофильные остатки размещены как 6 и 9 аминокислоты всех мотивов, структура белка может разрушиться. Поэтому было решено уменьшить свойство агрегации PPR, используя гидрофильные аминокислоты (аспарагин, аспарагиновую кислоту, глутамин, глутаминовую кислоту, лизин, аргинин, серин и треонин) в качестве 6 аминокислоты, предпочтительно 6 и 9 аминокислот, только в первом мотиве.
Конкретная процедура выглядит следующим образом.
В первом мотиве PPR (M1) от N-конца белка, способного связываться с нуклеиновой кислотой-мишенью, имеющей специфическую нуклеотидную последовательность:
(1) гидрофильную аминокислоту используют в качестве аминокислоты A6, предпочтительно, аспарагин или аспарагиновую кислоту используют в качестве аминокислоты A6, и
(2) дополнительно, в качестве аминокислоты A9 используют гидрофильную аминокислоту или глицин, предпочтительно, глутамин, глутаминовую кислоту, лизин или глицин, или
(3) аминокислота A6 и аминокислота A9 образованы любой из следующих комбинаций;
- комбинация аспарагина в качестве аминокислоты A6 и глутаминовой кислоты в качестве аминокислоты A9,
- комбинация аспарагина в качестве аминокислоты A6 и глутамина в качестве аминокислоты A9,
- комбинация аспарагина в качестве аминокислоты A6 и лизина в качестве аминокислоты A9, и
- комбинация аспарагиновой кислоты в качестве аминокислоты A6 и глицина в качестве аминокислоты A9.
Среди таких мотивов PPR особенно предпочтительны следующие:
(1st_A-1) мотив PPR, состоящий из последовательности SEQ ID NO: 402, имеющей такие аминокислотные замены в положениях 6 и 9, которые удовлетворяет любая из комбинаций, определенных ниже;
(1st_A-2) мотив PPR, содержащий последовательность (1st_A-1), имеющую замену, делецию или добавление от 1 до 9 аминокислот, кроме аминокислот в положениях 1, 4, 6, 9 и 34, и обладающий свойством связывания аденина;
(1st_A-3) мотив PPR, имеющий идентичность последовательности, по меньшей мере, 80% с последовательностью (1st_A-1), при условии, что аминокислоты в положениях 1, 4, 6, 9 и 34 идентичны, и обладающий свойством связывания аденина;
(1st_С-1) мотив PPR, состоящий из последовательности SEQ ID NO: 403;
(1st_C-2) мотив PPR, содержащий последовательность (1st_C-1), имеющую замену, делецию или добавление от 1 до 9 аминокислот, кроме аминокислот в положениях 1, 4, 6, 9 и 34, и обладающий свойством связывания цитозина;
(1st_C-3) мотив PPR, имеющий идентичность последовательности, по меньшей мере, 80% с последовательностью (1st_C-1), при условии, что аминокислоты в положениях 1, 4, 6, 9 и 34 идентичны, и имеющий свойство связывания цитозина;
(1st_G-1) мотив PPR, состоящий из последовательности SEQ ID NO: 404, имеющей такие аминокислотные замены в положениях 6 и 9, которые удовлетворяет любая из комбинаций, определенных ниже;
(1st_G-2) мотив PPR, содержащий последовательность (1st_G-1), имеющую замену, делецию или добавление от 1 до 9 аминокислот, кроме аминокислот в положениях 1, 4, 6, 9 и 34, и обладающий свойством связывания гуанина;
(1st_G-3) мотив PPR, имеющий идентичность последовательности, по меньшей мере, 80% с последовательностью (1st_G-1), при условии, что аминокислоты в положениях 1, 4, 6, 9 и 34 идентичны, и обладающий свойством связывания гуанина;
(1-й_U-1) мотив PPR, состоящий из последовательности SEQ ID NO: 405, имеющей такие аминокислотные замены в положениях 6 и 9, которые удовлетворяет любая из комбинаций, определенных ниже;
(1st_U-2) мотив PPR, содержащий последовательность (1st_U-1), имеющую замену, делецию или добавление от 1 до 9 аминокислот, кроме аминокислот в положениях 1, 4, 6, 9 и 34, и обладающий свойством связывания урацила; и
(1st_U-3) мотив PPR, имеющий идентичность последовательности, по меньшей мере, 80% с последовательностью (1st_U-1), при условии, что аминокислоты в положениях 1, 4, 6, 9 и 34 идентичны, и обладающий свойством связывания урацила:
- комбинация аспарагина в качестве аминокислоты в положении 6 и глутаминовой кислоты в качестве аминокислоты в положении 9,
- комбинация аспарагина в качестве аминокислоты в положении 6 и глутамина в качестве аминокислоты в положении 9,
- комбинация аспарагина в качестве аминокислоты в положении 6 и лизина в качестве аминокислоты в положении 9,
- комбинация аспарагиновой кислоты в качестве аминокислоты в положении 6 и глицина в качестве аминокислоты в положении 9.
На фиг. 6А показаны аминокислотные последовательности мотивов v3.2, а также последовательности мотивов v3.1. В v3.2 первый мотив выбран из 1st_A (SEQ ID NO: 402), 1st_C (SEQ ID NO: 403), 1st_G (SEQ ID NO: 404) и 1st_U (SEQ ID NO: 405), и второй и следующие мотивы выбраны из v2_C, v2_G, v2_U и v3.1_A. Использование любого из v3.2 в качестве первого мотива PPR от N-конца в белке PPR может улучшить свойство внутриклеточной агрегации.
(Другие)
Термин «идентичность», используемый в настоящем изобретении для нуклеотидной последовательности (также называемой нуклеотидной последовательностью) или аминокислотной последовательности, означает процентное соотношение числа совпадающих оснований или аминокислот, общих для двух последовательностей, выровненных оптимальным образом, если не указано иное. Другими словами, идентичность может быть вычислена в соответствии с уравнением: Идентичность=(Количество совпадающих положений/Общее количество положений) x 100, и ее можно вычислить с использованием имеющихся в продаже алгоритмов. Такие алгоритмы также включены в программы NBLAST и XBLAST, описанные в Altschul et al., J. Mol. Biol., 215 (1990) 403-410. Более подробно, поиск и анализ идентичности нуклеотидных или аминокислотных последовательностей можно выполнять с помощью алгоритмов или программ, хорошо известных специалистам в данной области техники (например, BLASTN, BLASTP, BLASTX и ClustalW). В случае использования программы, параметры могут быть надлежащим образом установлены специалистами в данной области техники, или также могут использоваться параметры по умолчанию для каждой программы. Конкретные процедуры этих способов анализа также хорошо известны специалистам в данной области техники.
В этом описании, когда идентичность выражена в долях для нуклеотидной последовательности или аминокислотной последовательности, более высокое процентное значение идентичности является предпочтительным в обоих случаях, если не указано иное, в частности, 70% или выше является предпочтительной, 80% или выше является более предпочтительной, 85% или выше является еще более предпочтительной, 90% или выше является еще более предпочтительной, 95% или выше является еще более предпочтительной и 97,5% или выше является еще более предпочтительной.
Что касается термина «последовательность, имеющая замену, делецию или добавление», используемого в настоящем изобретении в отношении мотива или белка PPR, количество замененных аминокислот или подобных конкретно не ограничивается каким-либо мотивом или белком, при условии, что мотив или белок, содержащий аминокислотную последовательность, выполняет желаемую функцию, если не указано иное. Количество заменяемых аминокислот или подобных может составлять от 1 до 9 или от 1 до 4, или даже большее количество аминокислот может быть заменено или подобное, если они заменены аминокислотами, имеющими аналогичные свойства. Способы получения полинуклеотидов или белков для таких аминокислотных последовательностей хорошо известны специалистам в данной области техники.
Аминокислоты, имеющие аналогичные свойства, относятся к аминокислотам с аналогичными физическими свойствами, таким как гидрофобность, заряд, рКа и растворимость, и относятся, например, к такой аминокислоте, как упомянуто ниже.
Гидрофобные (не полярные) аминокислоты: аланин, валин, глицин, изолейцин, лейцин, фенилаланин, пролин, триптофан, тирозин.
Не гидрофобные аминокислоты: аргинин, аспарагин, аспарагиновая кислота, глутаминовая кислота, глутамин, лизин, серин, треонин, цистеин, гистидин, метионин.
Гидрофильные аминокислоты: аргинин, аспарагин, аспарагиновая кислота, глутаминовая кислота, глутамин, лизин, серин, треонин.
Кислые аминокислоты: аспарагиновая кислота, глутаминовая кислота.
Основные аминокислоты: лизин, аргинин, гистидин.
Нейтральные аминокислоты: аланин, аспарагин, цистеин, глутамин, глицин, изолейцин, лейцин, метионин, фенилаланин, пролин, серин, треонин, триптофан, тирозин, валин.
Серосодержащие аминокислоты: метионин, цистеин.
Аминокислоты, содержащие ароматическое кольцо: тирозин, триптофан, фенилаланин.
Мотив PPR, белок, содержащий его, или нуклеиновые кислоты, кодирующие его по настоящему изобретению, могут быть получены специалистами в данной области техники с использованием обычных методик.
[Эффективность новых мотивов PPR]
(Связывающая способность)
Белки PPR, полученные с использованием новых мотивов PPR по настоящему изобретению (SEQ ID NOS: 9-12), не только подходят для получения белков PPR для относительно длинных РНК-мишеней, но также могут иметь более высокую эффективность связывания РНК по сравнению с белками PPR, полученными с использованием существующих мотивов PPR (SEQ ID NOS: 13-16) для той же РНК-мишени.
Другими словами, использование новых мотивов PPR по настоящему изобретению в белке PPR может увеличить способность связывания с РНК-мишенью по сравнению с использованием существующих мотивов PPR. Увеличивая связывающую способность, можно повысить эффективность манипуляции с РНК с использованием белка PPR в клетке. Например, эффективность внутриклеточного сплайсинга можно повысить, используя белок PPR, демонстрирующий высокую способность связывания с мишенью (см. пример 5).
Считается, что степень улучшения связывающей способности варьируется в зависимости от последовательности и длины мишени, и связывающая способность может быть увеличена, например, в 1,1 раза или более, более конкретно, в 1,3 раза или более, 2,0 раза или более, 3,0 раза или более или в 3,6 раза или более.
Связывающую способность с последовательностью-мишенью можно оценить с помощью EMSA (анализа изменения электрофоретической подвижности) или способа с использованием Biacore. EMSA является способом, использующим такое свойство нуклеиновой кислоты, что когда образец, состоящий из нуклеиновой кислоты, связанной с белком, подвергается электрофорезу, подвижность молекулы нуклеиновой кислоты изменяется по сравнению с подвижностью не связанной нуклеиновой кислоты. Анализаторы молекулярного взаимодействия, такие как Biacore в качестве типового примера, позволяют проводить кинетический анализ и, следовательно, позволяют проводить подробный анализ связывания белка с нуклеиновой кислотой.
Связывающую способность с последовательностью-мишенью также можно оценить с помощью RPB-ELISA, описанного ниже. В RPB-ELISA, значение, полученное вычитанием фонового сигнала (значение сигнала люминесценции, полученное с целевым белком PPR без добавления РНК-мишени) из люминесценции, полученной с образцом, содержащим целевой белок PPR и его РНК-мишень, можно использовать в качестве показателя связывающей способности целевого белка PPR и его РНК-мишени.
(Специфичность)
Белок PPR, полученный с использованием новых мотивов PPR по настоящему изобретению, может иметь более высокий потенциал специфичности к последовательности-мишени по сравнению с белком PPR, полученным с использованием существующих мотивов PPR для той же РНК-мишени.
То есть, используя новые мотивы PPR по настоящему изобретению в белке PPR, можно повысить специфичность для РНК-мишени по сравнению со случаем использования существующих мотивов PPR. Используя белок PPR, имеющий более высокую специфичность для РНК-мишени, можно избежать непреднамеренных эффектов в результате связывания с непреднамеренной РНК, когда РНК-мишенью манипулируют в клетке с использованием белка PPR.
Аффинность к последовательности-мишени может быть оценена специалистами в данной области с применением обычных способов. В RPB-ELISA, путем конструирования соответствующей РНК вне мишени для целевого белка PPR и определения связывающей способности для него (значение сигнала люминесценции) таким же образом, значение сигнала связывания для последовательности-мишени/значение сигнала связывания для последовательности вне мишени (S/N) может быть определено как индекс специфичности (аффинности) для РНК-мишени.
(Значение Kd)
Белок PPR, полученный с использованием новых мотивов PPR по настоящему изобретению, может иметь высокую аффинность (равновесную константу диссоциации, значение Kd) к РНК-мишени.
Значения Kd для последовательности-мишени можно рассчитать с помощью существующих способов, таких как EMSA. Значение Kd, используемое в настоящем изобретении, относится к значению, измеренному EMSA в условиях, описанных в разделе примеров, описанных ниже, если не указано иное.
Хотя значение Kd белка PPR, полученного с использованием новых мотивов PPR по настоящему изобретению, можно рассматривать как зависящее от последовательности и длины мишени, оно может составлять 10-7 M или меньше, 10-8 M или меньше, или порядка 10-9 M, когда длина последовательности-мишени составляет 18 оснований или больше. Согласно исследованию авторов настоящего изобретения, когда длина последовательности-мишени составляет 18 оснований, минимальное значение Kd (высокое сродство) составляет 1,95 × 10-9 в условиях примеров, что ниже, чем любое из ранее сообщенных значений Kd сконструированных белков PPR (см. таблицу 1). Кстати, было определено, что значения Kd коррелируют со значениями сигнала, полученными в экспериментах по связыванию RPB-ELISA. Можно оценить, что когда значение люминесценции RPB-ELISA (в соответствии с условиями, описанными в разделе «Примеры») составляет от 1 до 2×107, значение Kd составляет от 10-6 до 10-7 M, когда значение люминесценции RPB-ELISA составляет от 2 до 4×107, значение Kd составляет от 10-7 до 10-8 M, и когда значение люминесценции RPB-ELISA больше 4×107, значение Kd составляет 10-8 или меньше.
(Эффективность конструирования белка PPR)
Используя новые мотивы PPR по настоящему изобретению, можно эффективно сконструировать желаемый белок PPR. Эффективность конструирования можно рассчитать, определив долю успешного конструирования белков PPR с высокими значениями Kd с использованием существующих способов. Эффективность конструирования также можно рассчитать, используя значение сигнала люминесценции, полученное с помощью RPB-ELISA, вместо значения Kd таким же образом, как описано выше.
В частности, когда длина последовательности-мишени составляет 18 оснований, при использовании нового мотива PPR по настоящему изобретению, белки PPR со значением Kd 10-6 M или ниже (значение RPB-ELISA составляет 1×107 или выше) могут быть получены с эффективностью 50% или выше, более конкретно, 60% или выше, еще более конкретно, 70% или выше, еще более конкретно, 80% или выше. Согласно настоящему изобретению, белки PPR со значением Kd 10-7 M или ниже (значение RPB-ELISA составляет 2×107 или выше) для последовательности-мишени длиной 18 оснований могут быть получены с эффективностью 50% или выше, в частности, 55% или выше, более конкретно, 65% или выше, еще более конкретно, 75% или выше. Кроме того, согласно настоящему изобретению, белки PPR со значением Kd 10-8 M или ниже (значение RPB-ELISA составляет 4×107 или выше) для последовательности-мишени длиной 18 оснований могут быть получены с эффективностью 20% или выше, конкретно, 25% или выше, более конкретно, 30% или выше, еще более конкретно, 35% или выше.
Эффективность конструирования можно рассчитать на основе отношения значения сигнала связывания с последовательностью-мишенью/значения сигнала связывания с последовательностью вне мишени (S/N) с использованием метода RPB-ELISA.
В частности, с использованием новых мотивов PPR по настоящему изобретению, белки PPR с S/N 10 или выше для последовательности-мишени длиной 18 оснований могут быть получены с эффективностью 50% или выше, более конкретно, 55% или выше, еще более конкретно, 65% или выше, еще более конкретно, 75% или выше. Согласно настоящему изобретению, белки PPR с S/N 100 или выше для последовательности-мишени длиной 18 оснований могут быть получены с эффективностью 15% или выше, в частности, 20% или выше, более конкретно, 25% или выше, более конкретно, 30% или выше.
[Бесшовное клонирование гена белка PPR с использованием библиотеки частей]
Настоящее изобретение также представляет способ получения гена, кодирующего белок, содержащий n мотивов PPR, которые могут связываться с нуклеиновой кислотой-мишенью, состоящей из последовательности длиной n оснований, который включает следующие стадии:
выбор m частей PPR, необходимых для получения целевого гена, из библиотеки, состоящей из, по меньшей мере, 20 x m частей PPR, которые состоят из m видов промежуточных векторов Dest-a, ..., которые сконструированы так, чтобы они могли последовательно связываться, и вставлены с, по меньшей мере, 20 видами полинуклеотидов, включая 4 типа, кодирующие мотивы PPR, которые обладают свойствами связывания аденина, цитозина, гуанина, урацила и тимина, соответственно, и 16 видов одних и тех же кодирующих продуктов связывания двух из PPR мотивов, соответственно; и
реакция Golden Gate выбранных m видов частей PPR вместе с частями вектора с получением вектора, в который вставлено m продуктов полинуклеотидного связывания. n является целым числом m или больше, и m x 2 или меньше. n может быть, например, от 10 до 20.
В способе настоящего изобретения используют реакцию Golden Gate. В реакции Golden Gate, несколько фрагментов ДНК вставляют в вектор с использованием рестрикционного фермента типа IIS и ДНК-лигазы Т4. Фермент рестрикции типа IIS расщепляет нуклеиновую кислоту в положении за пределами последовательности распознавания, и поэтому когезионный конец может быть выбран свободно. Кроме того, поскольку он использует 4 основания, выступающие на конце для лигирования, он очень эффективен. Кроме того, последовательность распознавания не остается в конструкции, полученной после отжига и лигирования. Следовательно, полинуклеотиды, кодирующие мотивы PPR, могут быть легко лигированы (рис. 5). Особенно предпочтительным примером фермента рестрикции типа IIS является BsaI.
Способ по настоящему изобретению позволяет эффективное получение гена за счет использования библиотеки частей, должным образом разработанной с учетом характеристик белков PPR и реакции Golden Gate, даже если ген содержит большое количество повторяющихся последовательностей. Следовательно, этот способ полезен для получения гена белка, содержащего 15 или более мотивов PPR, которые могут связываться с нуклеиновой кислотой-мишенью длиной 15 оснований или более, что требует большего количества повторяющихся последовательностей. Когда m равно 10, путем выбора 10 частей PPR, необходимых для получения гена-мишени из библиотеки, состоящей из 200 частей PPR, при желании можно получить ген, кодирующий белок, содержащий от 10 до 20 мотивов PPR. В нижеследующих описаниях, объяснения могут быть сделаны на примере получения гена, кодирующего РНК-связывающий белок PPR, последовательность-мишень которого имеет длину от 10 до 20 оснований. Однако этот способ также можно применять для получения белка PPR для последовательности-мишени другой длины, а также его можно применять для получения ДНК-связывающего белка PPR.
В способе настоящего изобретения получают и используют библиотеку частей, содержащих одну или две последовательности, кодирующие мотив PPR (стадия 1 и стадия 2 на фиг. 5). Библиотеку частей можно получить, например, вставив последовательности мотивов PPR в 10 различных промежуточных векторов Dest-a, b, c, d, e, f, g, h, i и j. Промежуточные векторы сконструированы таким образом, что Dest-a - Dest-j последовательно и беспрепятственно лигируется реакцией Golden Gate. Последовательности мотива PPR, которые должны быть вставлены, могут состоять, по меньшей мере, из 20 видов последовательностей, включая 4 вида, кодирующих каждое основание (A, C, G и U), и 16 видов, кодирующих каждый из продуктов лигирования двух мотивов PPR (AA, AC, AG, AU, CA, CC, CG, CU, GA, GC, GG, GU, UA, UC, UG и UU). В этом случае библиотека частей включает, по меньшей мере, 200 типов частей.
Затем выбирают необходимые части в соответствии с нуклеотидной последовательностью-мишенью. В частности, например, каждая часть выбирается из каждой из библиотек частей Dest-a, b, c, d, e, f, g, h, i и j и подвергается реакции Golden Gate вместе с векторными частями (стадия 3 на фиг. 5). Если для всех промежуточных векторов выбирают промежуточный вектор, содержащий 1 мотив, лигируют 10 последовательностей, или, если используют промежуточные векторы, содержащие 2 мотива, лигируют 20 последовательностей. Когда желательно связать от 11 до 19 последовательностей, можно выбрать 1 мотив из каждой из любых библиотек Dest-x.
Части вектора, которые будут использоваться на стадии 3, могут быть выбраны из трех типов векторов CAP-x (необходимо учитывать ii аминокислоту в мотиве PPR, ближайшую к С-концу, но ii аминокислоты гуанин-связывающего мотива PPR и урацил-связывающего мотива PPR идентичны, см. не патентный документ 1, упомянутый выше). Если основанием, распознаваемым мотивом, ближайшим к С-концу, является аденин, можно использовать CAP-A, если это цитозин, можно использовать CAP-C, и если это гуанин или урацил, можно использовать CAP-GU.
Полученные плазмиды могут быть трансформированы в E. coli, затем амплифицированы и экстрагированы.
[Способ определения или анализа белка PPR]
Настоящее изобретение представляет способ определения или количественной оценки белка, содержащего n мотивов PPR, которые могут связываться с нуклеиновой кислотой-мишенью, состоящей из последовательности длиной n оснований, который включает следующие стадии:
стадия добавления раствора, содержащего кандидатный белок, к твердофазной нуклеиновой кислоте-мишени и определения или количественного определения белка, который связывается с нуклеиновой кислотой-мишенью.
Этот способ определения или анализа по настоящему изобретению полезен в качестве высокопроизводительного способа оценки эффективности связывания белков PPR.
Поскольку способ определения или анализа по настоящему изобретению основан на применении ELISA (ферментного иммуносорбентного анализа) (фиг. 7A), его можно назвать способом RPB-ELISA (ELISA связывания РНК-белка). Хотя способ описан в данном документе как способ оценки РНК-связывающих белков PPR, он также может применяться для оценки эффективности связывания ДНК-связывающих белков PPR с ДНК-мишенью.
Стадия добавления раствора, содержащего кандидатный белок, к твердофазной нуклеиновой кислоте-мишени может быть специфически проведена путем пропускания раствора, содержащего целевой связывающий белок на молекуле нуклеиновой кислоты-мишени, иммобилизованной на планшете. Иммобилизация молекулы нуклеиновой кислоты-мишени может быть достигнута с помощью различных существующих способов иммобилизации, например, предоставлением зонда нуклеиновой кислоты, содержащего модифицированную биотином молекулу нуклеиновой кислоты-мишени, в лунки планшета, покрытого стрептавидином.
С другой стороны, кандидатный белок, который необходимо измерить, может быть слит с маркерным белком, например, ферментом, таким как люцифераза, или флуоресцентным белком. Слияние с маркерным белком упрощает определение и количественную оценку.
Преимущество способа RPB-ELISA состоит в том, что он не требует специального оборудования, например, как Biacore. Кроме того, способ RPB-ELISA обеспечивает высокую производительность и позволяет за короткое время оценить связывание между белком и нуклеиновой кислотой. Кроме того, способ RPB-ELISA имеет то преимущество, что он обеспечивает достаточное определение при концентрации белка 6,25 нМ или выше в условиях, используемых в примерах, и аналогичным образом позволяет определение также с лизатами E. coli, и, следовательно, не требует очистки белка, связывающего нуклеиновую кислоту-мишень.
[Использование белка PPR]
(Комплексный и гибридный белок)
Мотив PPR или белок PPR, представленный в настоящем изобретении, можно превратить в комплекс путем связывания функциональной области. Мотив PPR или белок PPR также может быть связан с белковой функциональной областью с образованием слитого белка. Функциональная область относится к части, выполняющей такую функцию, как конкретная биологическая функция, проявляемая в живом организме или клетке, например ферментативная функция, каталитическая функция, ингибирующая функция, промоторная функция и т.д., или функцию маркера. Такая область состоит, например, из белка, пептида, нуклеиновой кислоты, физиологически активного вещества или лекарственного средства. В следующих пояснениях, комплекс по настоящему изобретению может быть объяснен со ссылкой на слитый белок в качестве примера, но специалисты в данной области техники могут также понимать комплексы, отличные от слитого белка, согласно пояснениям.
В одном из предпочтительных вариантов осуществления, функциональной областью является рибонуклеаза (РНКаза). Примерами РНКазы являются РНКаза А (например, панкреатическая рибонуклеаза А крупного рогатого скота, PDB 2AAS) и РНКаза Н.
В одном из предпочтительных вариантов осуществления, функциональной областью является флуоресцентный белок. Примеры флуоресцентного белка включают mCherry, EGFP, GFP, Sirius, EBFP, ECFP, mTurquoise, TagCFP, AmCyan, mTFP1, MidoriishiCyan, CFP, TurboGFP, AcGFP, TagGFP, Azami-Green, ZsGreen, EmGFP, HyPer, TagYFP, EYFP, Venus, YFP, PhiYFP, PhiYFP-m, TurboYFP, ZsYellow, mBanana, KusabiraOrange, mOrange, TurboRFP, DsRed-Express, DsRed2, TagRFP, DsRed-Monomer, AsRed2, mStrawberry, TurboFP602, mRFP1, JRed, KillerRed, HcRed, KeimaRed, mRasberry, mPlum, PS-CFP, Dendra2, Kaede, EosFP и KikumeGR. Предпочтительным примером является mClover3 с точки зрения улучшения агрегации и/или эффективной локализации в ядрах в качестве слитого белка.
В одном из предпочтительных вариантов осуществления, когда мишенью является мРНК, функциональной областью является функциональный домен, который увеличивает количество экспрессии белка из мРНК-мишени (WO2017/209122). Функциональным доменом, который увеличивает количество экспрессии белка из мРНК, может быть, например, весь или функциональная часть функционального домена белка, который, как известно, прямо или косвенно способствует трансляции мРНК. Более конкретно, это может быть домен, который направляет рибосомы к мРНК, домен, ассоциированный с иницииацией или стимулированием трансляции мРНК, домен, ассоциированный с транспортом мРНК из ядра, домен, ассоциированный со связыванием с мембраной эндоплазматического ретикулума, домен, содержащий сигнальную последовательность, удерживающую эндоплазматический ретикулум (ER), или домен, содержащий сигнальную последовательность эндоплазматического ретикулума. Более конкретно, домен, который направляет рибосомы на упомянутую выше мРНК, может быть доменом, содержащим весь или функциональную часть полипептида, выбранную из группы, состоящей из белка с регулируемой плотностью (DENR), последовательности 1, амплифицированной злокачественными Т-клетками (MCT-1), транскрипционно-контролируемого опухолевого белка (TPT1) и Lerepo4 (CCCH-домен цинкового пальца). Доменом, ассоциированным с инициацией трансляции или промотированием трансляции мРНК, упомянутой выше, может быть домен, содержащий весь или функциональную часть полипептида, выбранную из группы, состоящей из eIF4E и eIF4G. Доменом, ассоциированным с транспортировкой мРНК из ядра, упомянутым выше, может быть домен, содержащий весь или функциональную часть связывающего белка типа «петля на стебле» (SLBP). Доменом, ассоциированным со связыванием с мембраной эндоплазматического ретикулума, упомянутым выше, может быть домен, содержащий весь или функциональную часть полипептида, выбранного из группы, состоящей из SEC61B, ассоциированного с транслокацией белка альфа (TRAP-альфа), SR-альфа, Dia1 (цитохром b5 редуктаза 3) и p180. Вышеупомянутой сигнальной последовательностью, удерживающей эндоплазматический ретикулум (сигналом удержания ER), может быть сигнальная последовательность, содержащая последовательность KDEL (KEEL). Вышеупомянутой сигнальной последовательностью эндоплазматического ретикулума может быть сигнальная последовательность, содержащая MGWSCIILFLVATATGAHS.
В настоящем изобретении, функциональная область может быть слита с белком PPR на N-концевой стороне или C-концевой стороне или как на N-концевой стороне, так и на C-концевой стороне. Комплексный или слитый белок может включать множество функциональных областей (например, от 2 до 5). Кроме того, комплексный или слитый белок по настоящему изобретению может состоять из функциональной области и белка PPR, косвенно слитых через линкер или подобный.
(Нуклеиновая кислота, кодирующая белок PPR и т. д., вектор и клетку)
Настоящее изобретение также представляет нуклеиновую кислоту, кодирующую мотив PPR, белок PPR или слитый белок, упомянутые выше, и вектор, содержащий такую нуклеиновую кислоту (например, вектор для амплификации и вектор экспрессии). В качестве хозяина вектора для амплификации можно использовать E. coli или дрожжи. В этом описании вектор экспрессии означает вектор, содержащий, например, ДНК, имеющую промоторную последовательность, ДНК, кодирующую желаемый белок, и ДНК, имеющую терминирующую последовательность сверху, но они не обязательно должны быть расположены в этом порядке, пока выполняется желаемая функция. В настоящем изобретении могут использоваться рекомбинантные векторы, полученные с использованием различных векторов, которые обычно могут использоваться специалистами в данной области техники.
Белок PPR или слитый белок по настоящему изобретению может функционировать в эукариотических (например, животных, растительных, микробных (дрожжевых и т.д.) и простейших) клетках. Слитый белок по настоящему изобретению может функционировать, в частности, в клетках животных (in vitro или in vivo). Примеры животных клеток, в которые может быть введен белок PPR или слитый белок по настоящему изобретению, или вектор, экспрессирующий его, включают, например, клетки, полученные от людей, обезьян, свиней, коров, лошадей, собак, кошек, мышей и крыс. Примеры культивируемых клеток, в которые может быть введен белок PPR или слитый белок по настоящему изобретению или экспрессирующий его вектор, включают, например, клетки яичника китайского хомячка (СНО), клетки COS-1, клетки COS-7, клетки VERO (ATCC CCL-81), клетки BHK, клетки MDCK, полученные из почек собак, клетки хомячка AV-12-664, клетки HeLa, клетки WI38, клетки 293, клетки 293T и клетки PER.C6, но не ограничены ими.
(Применение)
С белком PPR или слитым белком по настоящему изобретению функциональная область может быть доставлена внутрь живого тела или клеток для функционирования специфическим к последовательности нуклеиновой кислоты образом. Комплекс, связанный с маркером, таким как GFP, может быть использован для визуализации желаемой РНК в живом организме.
С помощью белка PPR или слитого белка по настоящему изобретению, нуклеиновая кислота может быть модифицирована или разрушена специфическим к последовательности нуклеиновой кислоты образом внутри клеток или живых организмов, и может быть придана новая функция. В частности, РНК-связывающие белки PPR вовлечены во все этапы обработки РНК, найденные в органеллах, такие как расщепление, редактирование РНК, трансляция, сплайсинг и стабилизация РНК. Соответственно, такие применения способа, касающегося модификации белков PPR, предусмотренных настоящим изобретением, а также мотива PPR и белка PPR, представленных настоящим изобретением, как упомянуто ниже, можно ожидать в различных областях.
(1) Медицинское обслуживание
- Создание белка PPR, который распознает и связывается с определенной РНК, ассоциированный с конкретным заболеванием. Анализ последовательности-мишени и ассоциированных белков для конкретной РНК. Результаты анализа можно использовать для идентификации соединений для лечения заболевания.
Например, известно, что у животных аномалии белка PPR, идентифицированные как LRPPRC, вызывают синдром Лея франко-канадского типа (LSFC, синдром Лея, подострая некротизирующая энцефаломиелопатия). Настоящее изобретение может способствовать лечению (профилактике, терапевтическому лечению или ингибированию прогрессирования) LSFC. Многие из существующих белков PPR работают для определения сайтов редакции для манипуляций с РНК (преобразование генетической информации о РНК, часто C в U). Белки PPR этого типа имеют дополнительный мотив, который, как предполагается, взаимодействует с ферментами редактирования РНК на С-конце. Ожидается, что белки PPR, имеющие такую структуру, сделают возможным введение полиморфизма оснований или лечение заболевания или состояния, вызванного полиморфизмом оснований.
- Создание клеток с контролируемой репрессией/экспрессией РНК. Такие клетки включают стволовые клетки, состояние дифференциации или отсутствия дифференциации которых отслеживается (например, клетки iPS), клетки моделей для оценки косметических средств и клетки, в которых экспрессия функциональной РНК может быть включена или выключена с целью выяснения механизма действия и фармакологического тестирования для разработки лекарственных средств.
- Получение белка PPR, который специфически связывается с определенной РНК, ассоциированной с конкретным заболеванием. Такой белок PPR вводят в клетку с использованием плазмиды, вирусного вектора, мРНК или очищенного белка, и функция РНК, вызывающая заболевание, может быть изменена (улучшена) путем связывания белка PPR с РНК-мишенью в клетке. Примеры механизма изменения функции включают, например, изменение структуры РНК путем связывания, нокдаун путем разложения, изменение реакции сплайсинга путем сплайсинга, замену оснований и так далее.
(2) Сельское, лесное и рыбное хозяйство.
- Повышение урожайности и качества сельскохозяйственных культур, продуктов леса и морепродуктов.
- Скрещивание организмов с повышенной резистентностью к болезням, улучшенной устойчивостью к окружающей среде или улучшенными или новыми функциями.
Например, что касается гибридных культур растений первого поколения (F1), растение F1 может быть создано искусственно с использованием стабилизации митохондриальной РНК и контроля трансляции белками PPR, так что урожай и качество сельскохозяйственных культур могут быть улучшены. Манипуляции с РНК и редактирование генома с использованием белков PPR более точно и быстро позволяют улучшить разнообразие и селекцию (генетическое улучшение организмов) организмов по сравнению с традиционными методами. Кроме того, можно сказать, что манипуляции с РНК и редактирование генома с использованием белков PPR аналогичны классическим методам селекции, таким как селекция мутантов и обратное скрещивание, поскольку они не трансформируют признаки с чужеродным геном, как при генетической рекомбинации, но они являются методами с использованием РНК и геномов, изначально принадлежавших растениям и животным. Таким образом, они также могут уверенно и быстро справиться с глобальными продовольственными и экологическими проблемами.
(3) Химия
- Контроль количества экспрессии белка путем манипулирования ДНК и РНК при производстве полезных веществ с использованием микроорганизмов, культивируемых клеток, тел растений и животных (например, тел насекомых). Таким образом можно повысить производительность полезных веществ. Примерами полезных веществ являются белковые вещества, такие как антитела, вакцины и ферменты, а также относительно низкомолекулярные соединения, такие как фармацевтические промежуточные продукты, ароматизаторы и красители.
- Повышение эффективности производства биотоплива за счет модификации метаболических путей водорослей и микроорганизмов.
Примеры
[Пример 1: Разработка способа получения гена PPR]
(Дизайн мотива)
Сначала конструируют мотивы PPR. Для последовательностей мотивов PPR, используемых в искусственных белках PPR, о которых сообщалось до сих пор, используют консенсусные последовательности встречающихся в природе последовательностей мотивов PPR, экстрагированных различными способами. Среди них, белки PPR, полученные из последовательности мотива dPPR (не патентные документы 2, 3 и 6, упомянутые выше), имеют низкое значение Kd (высокое сродство). Эта последовательность мотива PPR в дальнейшем упоминается как мотив v1 PPR.
Что касается других последовательностей мотивов PPR, получают консенсусные последовательности с использованием только мотивов PPR, содержащих типовые комбинации 1, 4 и ii аминокислот, которые распознают каждое основание. В частности, типовыми комбинациями аминокислот, которые распознают каждое основание, являются: комбинация первого валина, четвертого треонина и ii аспарагина, которая распознает аденин, комбинация первого валина, четвертого аспарагина и ii серина, которая распознает цитозин, комбинация первого валина, четвертого треонина и ii аспарагиновой кислоты, которая распознает гуанин, и комбинация первого валина, четвертого аспарагина и ii аспарагиновой кислоты, которая распознает урацил, поэтому консенсусные аминокислотные последовательности экстрагируют из последовательностей мотива PPR, содержащих эти комбинации первой, четвертой и ii аминокислот, и эти последовательности используют в качестве последовательностей мотива PPR, которые специфически распознают аденин, цитозин, гуанин и урацил, соответственно (Фиг. 1-4, SEQ ID NOS: 9-12). Эти мотивы в дальнейшем называют мотивами v2 PPR. Такие же комбинации 1, 4 и ii аминокислот также используют для мотивов v1 PPR (SEQ ID NOS: 13-16).
(Бесшовное клонирование с использованием одно- и двухмотивных библиотек)
Разрабатывают способ клонирования для бесшовного лигирования этих сконструированных последовательностей мотивов PPR (фиг. 5). Клонирование выполняют в три стадии. На стадии 1 конструируют и получают последовательности мотивов. На стадии 2 получают библиотеки плазмид, в которых клонированы один или два мотива. На стадии 3 необходимое количество мотивов лигируют для завершения гена PPR-мишени.
Сначала получают плазмиды, в которых клонирована одна последовательность мотива PPR (номера от 4 до ii) (стадия 1). Плазмиды STEP 1 содержат последовательности мотива PPR, распознающие A, C, G и U, соответственно. На следующей стадии 2, фрагменты ДНК, содержащие последовательность мотива PPR в плазмидах стадии 1, клонируют в промежуточный вектор (Dest-x, его последовательность показана ниже). Плазмиды со стадии 1, которые позволяют вставлять один мотив, обозначают как P1a-vx-X, и в плазмидах, которые позволяют вставлять два мотива, находящиеся на стороне N-конца обозначают как P2a-vx-X, и находящиеся на стороне C-конца обозначают P2b-vx-X (vx является v1 или v2, и X является A, C, G или U). Для клонирования в Dest-х, сайт рестриктазы BsaI (BsaI рестриктаза распознает и расщепляет последовательность GGTCTCnXXXX (SEQ ID NO: 17), где ХХXX часть составляет выступающий конец с четырьмя основаниями (далее упоминается как маркерная последовательность). Последовательности, которые должны быть бесшовно лигированы, конструируют следующим образом.
Получают нуклеотидные последовательности, каждая из которых содержит последовательность мотива, и следующие последовательности, лигированные на 5' и 3' сторонах последовательности мотива:
ggtctcaatac (SEQ ID NO: 18) и gtggtgagacc (SEQ ID NO: 19) в случае P1a,
ggtctcaatac (SEQ ID NO: 18, указана выше) и gtggtcacatatgagacc (SEQ ID NO: 20) в случае P2a, или
ggtctcacatac (SEQ ID NO: 21) и gtggtgagacc (SEQ ID NO: 19, указана выше) в случае P2b,
методом генного синтеза, и их клонируют в pUC57-amp.
Имеется 10 типов Dest-x (Dest-a, b, c, d, e, f, g, h, i и j), и их последовательности конструируют таким образом, чтобы Dest-a - Dest-j можно было бесшовно лигировать в таком порядке.
Получают
gaagacataaactccgtggtcacATACagagaccaaggtctcaGTGGtcacatacatgtcttc (SEQ ID NO: 1) в качестве Dest-a,
gaagacatATACagagaccaaggtctcaGTGGtgacataatgtcttc (SEQ ID NO: 22) в качестве Dest-b,
gaagacatcATACagagaccaaggtctcaGTGGttacatatgtcttc (SEQ ID NO: 23) в качестве Dest-c,
gaagacatacATACagagaccaaggtctcaGTGGttacaatgtcttc (SEQ ID NO: 24) в качестве Dest-d,
gaagacattacATACagagaccaaggtctcaGTGGtgacatgtcttc (SEQ ID NO: 25) в качестве Dest-e,
gaagacattgacATACagagaccaaggtctcaGTGGttaatgtcttc (SEQ ID NO: 26) в качестве Dest-f,
gaagacatgttacATACagagaccaaggtctcaGTGGtcatgtcttc (SEQ ID NO: 27) в качестве Dest-g,
gaagacatggtcacATACagagaccaaggtctcaGTGGtatgtcttc (SEQ ID NO: 28) в качестве Dest-h,
gaagacattggttacATACagagaccaaggtctcaGTGGatgtcttc (SEQ ID NO: 29) в качестве Dest-i, and
gaagacatgtggtgacATACagagaccaaggtctcaGTGGtcttc (SEQ ID NO: 30) в качестве Dest-j
методом генного синтеза и клонируют в pUC57-kan.
Плазмиды, состоящие из каждого Dest-x, в который вставлены мотив PPR, соответствующий A, C, G или U, или два мотива PPR, которые распознают каждую из комбинаций оснований AA, AC, AG, AU, CA, CC, CG, CU, GA, GC, GG, GU, UA, UC, UG и UU, получают для всех Dest-x, чтобы подготовить библиотеки плазмид со стадии 1 для v1 и v2 (каждая включает 200 типов). В каждой из комбинаций, упомянутых выше, 40 нг только плазмиды P1a или 40 нг плазмиды P2a и 40 нг плазмиды P2b объединяют с 0,2 мкл 10х лигазного буфера (NEB, B0202S), 0,1 мкл BsaI (NEB, R0535S) и 0,1 мкл лигазы Quick (NEB, M2200S), и общий объем доводят до 1,9 мкл стерильной водой. Реакции проводят при 37°C в течение 5 минут и при 16°C в течение 5 минут, которые попеременно повторяют в течение 5 циклов в термоциклере (Biorad, 1861096J1). Далее добавляют 0,1 мкл 10х буфера Cut smart (NEB, B7204) и 0,1 мкл BsaI (NEB, R0535S), и реакции проводят при 37°C в течение 60 минут и 80°C в течение 10 минут. XL1-blue трансформируют 2,5 мкл реакционного раствора и отбирают в среде LB, содержащей 30 мкг/мл канамицина. Вставку желаемых последовательностей подтверждают секвенированием.
На стадии 3, Dest-a - Dest-j выбирают в соответствии с последовательностью-мишенью и клонируют в вектор CAP-x (не патентный документ 1, упомянутый выше). Если те, каждый из которых содержит один мотив, используют для всех промежуточных векторов, лигируют 10 мотивов, и если используют те, которые содержат по два мотива каждый, лигируют 20 мотивов. Плазмида, содержащая от 11 до 19 мотивов, может быть получена с использованием Dest-x, содержащего один мотив в любом положении. Например, когда получают последовательность PPR с 18 мотивами, используют Dest-a и Dest-b, содержащие один мотив, и другие плазмиды, содержащие два мотива.
Промежуточные векторы, используемые при клонировании на стадии 3, должны быть выбраны из трех типов векторов. Применяют CAP-A, когда нуклеотидом, который должен распознаваться мотивом, ближайшим к С-концу, является аденин; CAP-C, когда им является цитозин; и CAP-GU, когда им является гуанин или урацил. Их конструируют так, чтобы аминокислотная последовательность MGNSV (SEQ ID NO: 31) была добавлена на N-конце повтора PPR, и ELTYNTLISGLGKAGRARDPPV (SEQ ID NO: 32) была добавлена на стороне C-конца PPR повтора в результате клонирования их в промежуточные векторы для стадии 3.
Каждую из 10 видов промежуточных плазмид в количестве 20 нг, 1 мкл 10х лигазного буфера (NEB, B0202S), 0,5 мкл BpiL (Thermo, ER1012) и 0,5 мкл лигазы Quick (NEB, M2200S) объединяют, и конечный объем доводят до 10 мкл стерильной водой. Реакции проводят при 37°C в течение 5 минут и при 16°C в течение 7 минут в течение 15 циклов. Далее добавляют 0,4 мкл BpiL и реакции проводят при 37°C в течение 30 минут и 75°C в течение 6 минут. Затем добавляют 0,3 мкл 1 мМ АТФ и 0,15 мкл нуклеазы Plasmid safe nuclease (Epicenter, E3110K) и реакцию проводят при 37°C в течение 15 минут. E. coli (компетентные клетки штамма XL-1 Blue, Nippon Gene) трансформируют 3,5 мкл реакционного раствора и культивируют в среде LB, содержащей 100 мкг/мл спектиномицина при 37°C в течение 16 часов для селекции. Часть образованных колоний используют для амплификации области вставленного гена с использованием праймеров pCR8_Fw: 5'-TTGATGCCTGGCAGTTCCCT-3' (SEQ ID NO: 33) и pCR8_Rv: 5'-CGAACCGAACAGGCTTATGT-3' (SEQ ID NO: 34). В 0,2 мл пробирку добавляют 5 мкл 2x Go-метки (Promega, M7123), 1,5 мкл 10 мкМ pCR8_Fw, 1,5 мкл 10 мкМ pCR8_Rv и 2 мкл стерильной воды, и реакцию проводят при 98°C в течение 2 минут, затем в течение 15 циклов при 98°C в течение 5 секунд, 55°C в течение 10 секунд и 72°C в течение 2,5 минут в термоциклере для проведения реакции амплификации ДНК. Часть реакционного раствора подвергают электрофорезу с использованием MultiNA (SHIMADZU, MCE202) для подтверждения размера вставленного фрагмента ДНК. Используя мотивы v1 и v2, получают три клона для каждого из трех типов белков PPR с 18 мотивами (PPR1, PPR2 и PPR3, SEQ ID NOS: 35-37 и 40-42) (v1_PPR1, v1_PPR2, v1_PPR3, v2_PPR1, v2_PPR2 и v2_PPR3), результаты которых показаны на фиг. 6B. Для v1 получают полосы правильного размера, за исключением второго клона PPR2. Для v2 для всех клонов получают полосы правильного размера. Кроме того, их последовательности подтверждают секвенированием. Эти результаты показывают, что гены белка PPR могут быть эффективно сконструированы путем клонирования в соответствии с этим способом.
[Пример 2: Конструирование высокопроизводительной системы оценки эффективности связывания для РНК-связывающего белка]
В общем, оценку связывания между связывающим нуклеиновую кислоту белком и молекулой нуклеиновой кислоты проводят способом с использованием EMSA или Biacore. EMSA (анализ изменения электрофоретической подвижности) является способом, использующим свойство, заключающееся в том, что при электрофорезе связанных вместе образца белка и нуклеиновой кислоты подвижность молекулы нуклеиновой кислоты изменяется по сравнению с подвижностью не связанной молекулы. Этот способ имеет недостатки в том, что для него требуется очищенный белок, операции являются сложными и невозможно одновременно анализировать большое количество образцов. Анализаторы молекулярного взаимодействия, типовым примером которых является Biacore, позволяют анализировать кинетику реакции и, следовательно, позволяют проводить подробный анализ связывания белка с нуклеиновой кислотой. Однако для них также требуется очищенный белок и специальное оборудование. Поэтому изобретатели продумали способ, который позволяет оценить связывание белок-нуклеиновая кислота за короткое время с высокой производительностью.
ELISA (ферментный иммуносорбентный анализ) обычно используют для анализа связывания антитела (белка) и белка. В этом способе первичное антитело фиксируется на планшете с лунками, в который добавляют раствор, содержащий определяемый белок, и после промывки добавляют вторичное антитело, определяемое по развитию цвета или люминесценции, и количественно определяют для определения количества оставшегося белка в качестве объекта анализа. Применяя этот способ, изобретатели разработали систему, в которой молекулу нуклеиновой кислоты фиксируют на планшете, раствор, содержащий целевой белок, связывающий нуклеиновую кислоту, выливают в планшет, и количественно определяют количество связанного белка (рис. 7А). Анализируемую нуклеиновую кислоту фиксируют путем добавления зонда нуклеиновой кислоты, состоящего из нуклеиновой кислоты, имеющей модифицированный биотином конец, в лунки планшета, покрытые стрептавидином. За счет слияния анализируемого белка, связывающего нуклеиновую кислоту, с люциферазой или флуоресцентным белком определение может быть упрощено. Кроме того, измеряемый белок необязательно может быть очищен, и анализ может быть выполнен с использованием неочищенного экстракта клеток, в котором экспрессируется измеряемый белок, связывающий нуклеиновую кислоту (культивируемых клеток животных, дрожжей, E. coli и т. д.), и тем самым можно сократить время очистки (рис. 7B). Этот способ, используемый для измерения связывания РНК и РНК-связывающего белка, в дальнейшем называется RPB-ELISA (ELISA связывания РНК-белок).
Для создания экспериментальной системы получают рекомбинантный белок MS2 и связывающийся с ним РНК-зонд. Ген белка MS2, слитый с белком люциферазы на N-конце и 6х гистидиновой меткой на C-конце, получают путем синтеза гена и клонируют в вектор pET21b (NL_MS 2_HIS, SEQ ID NO: 357). В качестве РНК зонда синтезируют последовательность-мишень, содержащую связывающую последовательность MS2 (RNA_4, SEQ ID NO: 64), и последовательность вне мишени, не содержащую связывающую последовательность MS2 (RNA_51, SEQ ID NO: 247), каждая из которых имеет 5'-конец, модифицированный биотинилированием (Greiner). Штамм E. coli Rosetta (DE3) трансформируют плазмидой экспрессии белка MS2 и культивируют в течение ночи при 37°C в 2 мл среды LB, содержащей 100 мкг/мл ампициллина. Затем 2 мл культуральной среды добавляют к 300 мл среды LB, содержащей 100 мкг/мл ампициллина, и культивируют при 37°C до достижения OD600 0,5-0,8. После снижения температуры среды, содержащей культивируемые клетки, до 15°C, добавляют IPTG до конечной концентрации 0,1 мМ, и культивирование продолжают в течение еще 12 часов. Культуральную среду центрифугируют при 5000 x g и 4°C в течение 10 минут для сбора клеток, добавляют 5 мл буфера для лизиса (20 мМ Tris-HCl, pH 8,0, 150 мМ NaCl, 0,5% NP-40, 1 мМ DTT, 1 мМ EDTA), смесь перемешивают с помощью вихревой мешалки и клетки разрушают обработкой ультразвуком. Обработанную ультразвуком смесь центрифугируют при 15000 об/мин и 4°C в течение 10 минут и собирают супернатант. Половину супернатанта хранят при -80°C до использования в качестве лизата E.coli, и остальную часть очищают аффинной очисткой с использованием гистидиновой метки и Ni-NTA. Сначала 200 мкл гранул Ni-NTA агарозы (Qiagen, кат. № 30230) центрифугируют, и гранулы собирают. Гранулы уравновешивают добавлением 100 мкл промывочного буфера, и перемешивают смесь на ротаторе при 4°C в течение 1 часа. Весь объем уравновешенных гранул смешивают с раствором белка, и реакцию проводят при 4°C в течение 1 часа. Затем гранулы собирают центрифугированием при 2000 об/мин в течение 2 минут и промывают 10 мл промывочного буфера (20 мМ Tris-HCl, pH 8,0, 500 мМ NaCl, 0,5% NP-40, 10 мМ имидазола) для удаления факторов, которые неспецифически связаны с гранулами. Элюирование проводят 60 мкл буфера для элюирования (20 мМ Tris-HCl, pH 8,0, 500 мМ NaCl, 0,5% NP-40, 500 мМ имидазола). Степень очистки подтверждают SDS-PAGE. Элюат диализируют в течение ночи при 4°C против 20 мМ Tris-HCl, pH 8,0, 150 мМ NaCl, 0,5% NP-40, 1 мМ DTT, 1 мМ EDTA.
Измеряют люминесценцию люциферазы лизата E. coli и очищенного раствора белка MS2. В 96-луночный белый планшет добавляют 40 мкл субстрата люциферазы (Promega, E151A), разведенного 2500-кратно люминесцентным буфером (20 мМ Tris-HCl, pH 7,6, 150 мМ NaCl, 5 мМ MgCl2, 0,5% NP-40, 1 мМ DTT) и 40 мкл лизата E. coli или 40 мкл очищенного раствора белка MS2 и реакцию проводят в течение 5 минут, после чего люминесценцию измеряют с помощью планшетного ридера (PerkinElmer, 5103-35). На основании полученных люминесценций, их разбавляют буфером для лизиса (20 мМ Tris-HCl, pH 7,6, 150 мМ NaCl, 5 мМ MgCl2, 0,5% NP-40, 1 мМ DTT, 0,1% BSA) для получения продуктов разведения 0,01×108, 0,02×108, 0,09×108, 0,38×108, 1,50×108 и 6,00×108 ЛЕ/мкл.
В 96-луночный белый планшет, покрытый стрептавидином (Thermo fisher, 15502) добавляют 2,5 пмоль биотинилированного РНК зонда, реакцию проводят при комнатной температуре в течение 30 минут и планшет промывают буфером для лизиса. Для измерения фона также готовят лунки, в которые не добавляют биотинилированную РНК, но добавляют буфер для лизиса (-Зонд). Затем добавляют блокирующий буфер (20 мМ Tris-HCl, pH 7,6, 150 мМ NaCl, 5 мМ MgCl2, 0,5% NP-40, 1 мМ DTT, 1% BSA), и поверхность планшета блокируют при комнатной температуре в течение 30 минут. Затем в каждую лунку добавляют 100 мкл лизата E. coli или раствора очищенного белка, разведенного выше, и реакцию связывания проводят при комнатной температуре в течение 30 минут. Затем лунки промывают 5 раз 200 мкл промывочного буфера (20 мМ Tris-HCl, pH 7,6, 150 мМ NaCl, 5 мМ MgCl2, 0,5% NP-40, 1 мМ DTT). В каждую лунку добавляют 40 мкл субстрата люциферазы (Promega, E151A), разведенного 2500-кратно промывочным буфером, реакцию проводят в течение 5 минут, и затем измеряют люминесценцию с помощью планшетного ридера (PerkinElmer, 5103-35).
Фон (значение сигнала люминесценции, полученное с добавлением белка PPR и без добавления РНК) вычитают из люминесценций образцов, к которым добавлен раствор, содержащий каждый из РНК и белка MS2, и полученные значения используют в качестве связывающей способности между белком MS2 и РНК.
Результаты показаны на фиг. 7C. Специфическое связывание белка MS2 с РНК-мишенью (Посл.-мишень) определяют и для раствора очищенного белка (очищенный белок), и для лизата E. coli (лизат). Люминесценция 6,0×108 ЛЕ/мкл соответствует 100 нМ очищенного белка MS2, и поэтому было найдено, что определение может быть в достаточной степени достигнуто при концентрации белка 6,25 нМ (0,38×108 ЛЕ/мкл) или выше. Кроме того, определение также возможно с лизатом E. coli, и поэтому было найдено, что очистка белка не требуется.
[Пример 3: Эксперимент по сравнению эффективности связывания РНК для белков PPR с 18-мотивами, полученных с использованием существующих или новых последовательностей мотивов PPR]
Чтобы оценить эффективность связывания РНК белков PPR, полученных с использованием мотивов PPR v1 или v2, рекомбинантные белки получают в E.coli, и их эффективность связывания оценивают с помощью RPB-ELISA. Для сравнения определяют 5 видов последовательностей-мишеней (T_1, T_2, T_3, T_4 и T_5, SEQ ID NOS: 46-50), конструируют белки PPR, связывающиеся с каждой из них, и получают гены, кодирующие их (v1_PPR1, v1_PPR2, v1_PPR3, v1_PPR4, v1_PPR5, v2_PPR 1, v2_PPR2, v2_PPR3, v2_PPR4 и v2_PPR5, SEQ ID NOS: 35-39 и 40-45). К каждому полученному гену PPR добавляют ген белка люциферазы на N-конце, и последовательность his-tag добавляют на C-конце, и их клонируют в вектор pET21 (NL_v1_PPR1, NL_v1_PPR2, NL_v1_PPR3, NL_v1_PPR4, NL_v1_PPR5, NL_v2_PPR1, NL_v2_PPR2, NL_v2_PPR3, NL_v2_PPR4 и NL_v2_PPR5, SEQ ID NOS: 51-60). Штамм Rosetta (DE3) трансформируют плазмидами экспрессии PPR. E. coli культивируют в 2 мл LB среды, содержащей 100 мкг/мл ампициллина, при 37°С в течение 12 часов. Когда OD600 достигает 0,5-0,8, культуральную среду переносят в инкубатор при 15°C и выстаивают в течение 30 минут. Затем добавляют 100 мкл раствора IPTG (конечная концентрация IPTG, 0,1 мМ), и культивирование продолжают далее при 15°C в течение 16 часов. Дебрис E.coli собирают центрифугированием при 5000 х g и 4°C в течение 10 мин, к дебрису добавляют 1,5 мл буфера для лизиса (20 мМ Tris-HCl, рН 8,0, 150 мМ NaCl, 0,5% NP-40, 1 мМ MgCl2, 2 мг/мл лизозима, 1 мМ PMSF, 2 мкл 10 мг/мл ДНКазы), и смесь замораживают при -80°C в течение 20 минут. Клетки криоразрушают с проницаемостью при 25°C в течение 30 минут. Затем смесь разрушенных клеток центрифугируют при 3700 об/мин и 4°C в течение 15 минут и собирают супернатант, содержащий растворимый белок PPR (лизат E. coli).
Синтезируют РНК зонды, состоящие из сконструированной последовательности из 30 оснований, содержащей 18 оснований последовательности-мишени и модифицированной биотинилированием на 5' конце (RNA_1, RNA_2, RNA_3, RNA_4 и RNA_5, SEQ ID NOS: 61-65) (Grainer). В покрытый стрептавидином планшет (Thermo fisher) добавляют 5'-концевые биотинилированные РНК зонды, реакцию проводят при комнатной температуре в течение 30 минут, и планшет промывают буфером для лизиса (20 мМ Tris-HCl, pH 7,6, 150 мМ NaCl, 5 мМ MgCl2, 0,5% NP-40, 1 мМ DTT, 0,1% BSA). Для измерения фона также готовят лунки, в которые не добавляют РНК, но добавляют 100 мкл буфера для лизиса, 1 мкл 100 мМ DTT и 1 мкл 40 единиц/мкл ингибитора РНКазы (Takara, 2313A). Затем добавляют 200 мкл блокирующего буфера (20 мМ Tris-HCl, pH 7,6, 150 мМ NaCl, 5 мМ MgCl2, 0,5% NP-40, 1 мМ DTT, 1% BSA), и поверхность планшета блокируют при комнатной температуре в течение 30 минут. Затем в каждую лунку добавляют 100 мкл лизата E.coli, содержащего слитый с люциферазой белок PPR, имеющий уровень люминесценции 1,5 × 108 ЛЕ/мкл, и реакцию связывания проводят при комнатной температуре в течение 30 минут. Лунку промывают 5 раз 200 мкл промывочного буфера (20 мМ Tris-HCl, pH 7,6, 150 мМ NaCl, 5 мМ MgCl2, 0,5% NP-40, 1 мМ DTT). В каждую лунку добавляют 40 мкл субстрата люциферазы (Promega, E151A), разведенного 2500-кратно промывочным буфером, реакцию проводят в течение 5 минут, и затем измеряют люминесценцию с помощью планшетного ридера (PerkinElmer, 5103-35). Фон (значение сигнала люминесценции, полученное при добавлении белка PPR и без добавления РНК) вычитают из люминесценции образцов, к которым добавляют раствор, содержащий каждую РНК и белок PPR, и полученные значения используют в качестве связывающей способности между белком PPR и РНК.
Результаты показаны на фиг. 8. Все конструкции, полученные с последовательностью мотива v2, показали увеличение (от 1,3 до 3,6 раз) способности связывания с последовательностью-мишенью по сравнению с теми, которые были получены с последовательностью мотива v1. Кроме того, получают 2 вида РНК зондов, имеющих последовательность вне мишени (вне мишени 1 и вне мишени 2) (SEQ ID NOS: 66 и 69), и исследуют связывание белков с ними. В результате все конструкции, полученные с v2, показали более высокое соотношение сигнал связывания мишени/сигнал связывания вне мишени (S/N) по сравнению с теми, которые были получены с v1 (фиг. 8, вверху слева), и поэтому было определено, что v2 показывает более высокую аффинность и специфичность к мишени по сравнению с v1.
[Пример 4: Подробный анализ эффективности связывания РНК белков PPR, полученных с мотивом v2]
(Оценка специфичности)
Используя мотив v2, получают белки PPR для 23 видов последовательностей-мишеней (T_1 - T_3 и T_5 - T_24, SEQ ID NOS: 46-48 и 51-69) (NL_v2_PPR1-3 и NL_v2_PPR5-24, SEQ ID NOS: 56-58 и 70-88), и RPB-ELISA используют для анализа связывания всех комбинаций. Экспериментальный способ такой же, как используют в Примере 3.
Результаты показаны на фиг. 9 (верхняя часть). Было определено, что способность связывания с мишенью была самой сильной у 21 вида белков PPR, за исключением v2_17 и v2_24. Эти результаты показывают, что белки PPR могут быть стабильно получены с использованием мотива v2, и их специфичность связывания высока.
Используя мотив V3.1, белки PPR для тех же 23 видов последовательностей-мишеней получают аналогичным образом (SEQ ID NO: 411-43 для нуклеотидных последовательностей и SEQ ID NO: 434-456 для аминокислотных последовательностей), и связывания всех комбинаций анализируют с помощью RPB-ELISA. Экспериментальный способ такой же, как используют в Примере 3.
Результаты показаны на фиг. 9 (нижняя часть) и в таблице ниже. Получены конструкции, которые показали улучшенную связывающую способность по сравнению с V2.
[Таблица 9]
Характеристики связывания РНК белков PPR, показанные на фиг. 9, суммированы в приведенных ниже таблицах в виде числовых значений (значений log2).
[Таблица 10-1]
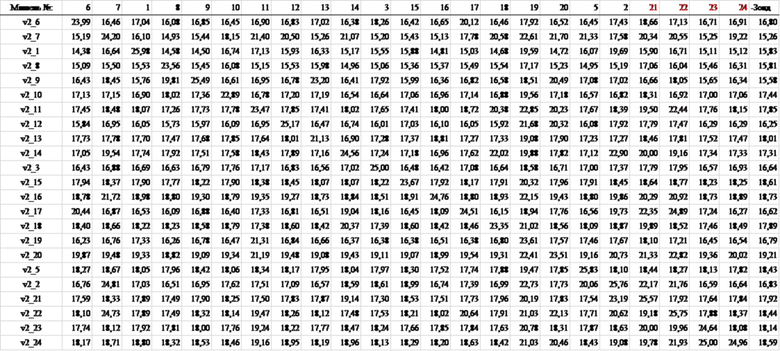
[Таблица 10-2]
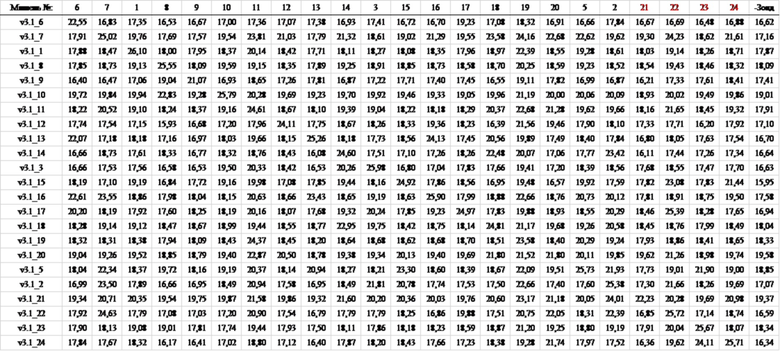
(Оценка аффинности)
Кроме того, EMSA выполняют для расчета аффинности (значение Kd) каждого белка PPR к его РНК-мишени. Плазмиды экспрессии E. coli конструируют для 10 видов белков PPR из 23 видов белков, упомянутых выше, к которым добавлена последовательность стрептавидин-связывающего пептида на стороне N-конца и последовательность 6x His-tag на стороне C-конца (SBP_v2_PPR1_HIS, SBP_v2_PPR2_HIS, SBP_v2_PPR3_HIS, SBP_v2_PPR6_HIS, SBP_v2_PPR9_HIS, SBP_v2_PPR12_HIS, SBP_v2_PPR15_HIS, SBP_v2_PPR16_HIS, SBP_v2_PPR20_HIS и SBP_v2_PPR24_HIS, SEQ ID NOS: 89-97). Штамм Rosetta (DE3) E. coli трансформируют плазмидами и культивируют в течение ночи при 37°C в 2 мл среды LB, содержащей 100 мкг/мл ампициллина. Затем 2 мл культуральной среды переносят в 300 мл среды LB, содержащей 100 мкг/мл ампициллина, и культивирование проводят при 37°C до тех пор, пока OD600 не достигнет 0,5-0,8. После культивирования температуру среды снижают до 15°C, затем добавляют 0,1 мМ IPTG и культивирование продолжают еще 12 часов. Культуральную среду центрифугируют при 5000 x g и 4°C в течение 10 минут для сбора клеток, добавляют 5 мл буфера для лизиса (20 мМ Tris-HCl, pH 8,0, 150 мМ NaCl, 0,5% NP-40, 1 мМ DTT, 1 мМ EDTA), смесь перемешивают на смесителе Voltex и обрабатывают ультразвуком для разрушения клеток. Разрушенную смесь центрифугируют при 15000 об/мин и 4°C в течение 10 минут и собирают супернатант.
Затем целевые белки очищают аффинной хроматографией с использованием метки SBP. Streptavidine Sepharose High Performance (GE Healthcare, 17511301) берут в объеме 100 мкл, гранулы собирают центрифугированием и уравновешивают промывочным буфером (20 мМ Tris-HCl, pH 8,0, 500 мМ NaCl, 0,5% NP-40). Уравновешенные гранулы осторожно смешивают с каждым из ранее собранных клеточных экстрактов и пермеабилизируют при 4°C в течение 10 минут. Весь объем смеси гранул загружают в колонку, и затем гранулы промывают 10 мл промывочного буфера. Элюирование проводят с помощью буфера для элюирования (20 мМ Tris-HCl, pH 8,0, 500 мМ NaCl, 2 мМ биотина).
Затем проводят аффинную очистку с использованием гистидиновой метки. Сначала собирают 200 мкл Ni-NTA агарозы (Qiagen, 30230) и после центрифугирования собирают гранулы. К гранулам добавляют 100 мкл промывочного буфера, и гранулы уравновешивают пермеабилизацией при 4°C в течение 1 часа. Весь объем уравновешенных гранул смешивают с раствором белка, элюированным с гранул SBP, и реакцию проводят при 4°C в течение 1 часа. Гранулы собирают центрифугированием при 2000 об/мин в течение 2 минут, и факторы, неспецифически связывающиеся с гранулами, удаляют с помощью 10 мл промывочного буфера (20 мМ Tris-HCl, pH 8,0, 500 мМ NaCl, 0,5% NP-40, 10 мМ имидазола). Элюирование проводят 60 мкл буфера для элюирования (20 мМ Tris-HCl, pH 8,0, 500 мМ NaCl, 0,5% NP-40, 500 мМ имидазол). Степень очистки подтверждают с помощью SDS-PAGE, и элюированный раствор диализируют в течение ночи при 4°C против 20 мМ Tris-HCl, pH 8,0, 150 мМ NaCl, 0,5% NP-40, 1 мМ DTT, 1 мМ EDTA.
Общее количество белка, полученного после диализа, оценивают с использованием набора для анализа белков Pierce 660nm Protein Assay Kit (Thermo fisher, 22662). Для определения количества целевого белка, каждый диализованный образец подвергают SDS-PAGE в 10% полиакриламидном геле и проводят окрашивание CBB. Изображение окрашенного геля было получают с помощью системы визуализации ChemiDoc Touch MP Imaging System (Biorad). Полную интенсивность полосы и интенсивность целевой полосы получают из изображения геля. Количество целевого белка рассчитывают путем умножения общего количества белка на отношение интенсивности целевой полосы к интенсивности общей полосы. Это значение используют для расчета молярной концентрации очищенного белка в диализованном образце. На основе рассчитанных выше молярных концентраций готовят разведенные растворы белка 400 нМ, 200 нМ, 100 нМ, 50 нМ, 20 нМ, 10 нМ, 5 нМ, 2 нМ и 1 нМ. Разведение проводят связывающим буфером (20 мМ Tris-HCl, pH 8,0, 150 мМ NaCl, 0,5% NP-40, 1 мМ DTT, 1 мМ EDTA). Конечные концентрации 5'-концевых биотинилированных РНК зондов (RNA_1, RNA_2, RNA_3, RNA_6, RNA_9, RNA_12, RNA_15, RNA_16, RNA_20 и RNA_24) доводят до 20 нМ с помощью связывающего буфера. Образцы РНК подвергают термообработке при 75°C в течение 1 минуты, гасят и используют в следующих экспериментах.
Раствор белка каждой концентрации, приготовленный выше, смешивают с 20 нМ раствором зонда РНК и проводят реакцию связывания при 25°C в течение 20 минут.
После реакции добавляют 2 мкл 80% глицерина, и смесь в достаточной степени суспендируют. Затем 10 мкл смеси наносят на 7,5% гель ATTO и проводят электрофорез при C.V. 150 В в течение 30 минут.
Гели после электрофореза переносят на мембрану Hybond N+ (GE, RPN203B). Затем РНК подвергают УФ перекрестному связыванию с мембраной с использованием ASTEC Dual UV Transilluminator UVA-15 (Astec, 49909-06). Мембрану блокируют блокирующим буфером (6,7 мМ NaH2PO4 · 2H2O, 6,7 мМ Na2HPO4 · 2H2O, 125 мМ NaCl, 5% SDS). В этой операции к блокирующему буферу заранее добавляют 0,5 мкл Стрептавидина-HRP (Abcam, ab7403) и проводят реакцию антиген-антитело с пермеабилизацией в течение 15 минут. Блокирующий буфер удаляют и добавляют 20 мл промывочного буфера (0,67 мМ NaH2PO4 · 2H2O, 0,67 мМ Na2HPO4 · 2H2O, 12,5 мМ NaCl, 0,5% SDS) для промывки мембраны. Эту процедуру промывки повторяют пять раз, и затем добавляют 20 мл уравновешивающего буфера (100 мМ Tris-HCl, pH 9,5, 100 мМ NaCl, 10 мМ MgCl2) для пермеабилизации мембраны в течение 5 минут. Затем к мембране добавляют субстрат Immunobilon Western Chemiluminescent HRP Substrate (Millipore, кат. № WBKLS0100) и определяют полосы с использованием хемилюминесценции биотинилированной РНК. Изображения геля получают с помощью системы визуализации ChemiDoc Touch MP Imaging System (Biorad). Рассчитывают интенсивности полос полосы несвязанного зонда РНК и полосы, сдвинутой за счет связывания с белком. Равновесную константу диссоциации (значение Kd) рассчитывают из молярной концентрации белка и соотношения соответствующей сдвинутой полосы в соответствии с уравнением Хилла.
Результаты показаны на фиг. 10. Было обнаружено, что полученные белки PPR имеют значение Kd от 10-9 до 10-7 M для мишеней. Минимальное значение (высокая аффинность) составляет 1,95 × 10-9, что является самым низким значением Kd среди значений Kd для сконструированных белков PPR, о которых сообщалось до сих пор (см. Таблицу 1). Также было определено, что эти значения Kd коррелируют со значениями сигнала, полученными в экспериментах по связыванию на основе RPB-ELISA (R2=-0,85). На основе этих результатов можно оценить, что значение Kd составляет от 10-6 до 10-7 M для значения люминесценции RPB-ELISA от 1 до 2×107, от 10-7 до 10-8 M для RPB-ELISA значение люминесценции от 2 до 4×107, и ~10-8 или ниже для значения люминесценции RPB-ELISA более 4×107.
(Оценка степени успешного конструирования)
Далее получают белки PPR для 72 видов последовательностей-мишеней (T1-T3 и T6-T76, SEQ ID NOS: 46-48, 51-69 и 117-168) с использованием мотива v2 (NL_v2_PPR1-3 и NL_v2_PPR6-76, SEQ ID NOS: 56-58, 70-88 и 169-220), и вероятность успешного конструирования рассчитывают с использованием RPB-ELISA. Получают биотинилированные РНК зонды, содержащие последовательность-мишень (РНК_1-3 и РНК_6-76, SEQ ID NOS: 61-63, 98-116 и 221-272), и биотинилированный РНК зонд (РНК51, SEQ ID NO: 247) содержащий последовательность вне мишени (T_51, SEQ ID NO: 143) (Greinar). Экспериментальный способ такой же, как и в примере 3. Результаты показаны на фиг. 11.
Из 72 видов белков PPR, 63 (88%) оценивают как имеющие значение Kd 10-6 M или ниже (значение RPB-ELISA 1×107 или выше), 57 (79%) оценивают как имеющие значение Kd 10-7 M или ниже (значение RPB-ELISA 2×107 или выше), и 43 (40%) оценивают как имеющие значение Kd 10-8 M или ниже (значение RPB-ELISA 4×107 или выше). Значение, полученное путем деления сигнала связывания с мишенью на сигнал связывания вне мишени (S/N), используют в качестве значения для оценки специфичности. 54 (75%) показали S/N выше 10, и 23 (32%) показали S/N выше 100. Эти результаты показывают, что специфичные к последовательности РНК-связывающие белки могут быть эффективно получены путем получения белков PPR с использованием мотива v2.
(Оценка активности связывания мишени в зависимости от количества мотивов PPR)
Проводят анализ зависимости количества мотивов PPR и активности связывания с мишенью. Определяют 13 видов последовательностей-мишеней из 18 оснований, и 3 нуклеотида или 6 нуклеотидов на 5'-конце каждой последовательности удаляют для конструирования последовательностей-мишеней из 15 оснований (T_1a, T_49a, T_3a, T_14a, T_40a, T_12a, T_13a, T_2a, T_38a, T_37a_T_39a, T_56a и T_68a, SEQ ID NOS: 273, 275, 277, 279, 281, 283, 285, 287, 289, 291, 293, 395 и 297) и последовательностей-мишеней из 12 оснований (T_1b, T_49b, T_3b, T_14b, T_40b, T_12b, T_13b, T_2b, T_38b, T_37b T_39b, T_56b и T_68b, SEQ ID NOS: 274, 276, 278, 280, 282, 284, 286, 288, 290, 292, 294, 296 и 298), соответственно. Получают соответствующие белки PPR (имеющие 15 мотивов названы PPRxa, и имеющие 12 мотивов названы PPRxb) (NL_v2_PPR1, 1a и 1b; NL_v2_PPR49, 49a и 49b; NL_v2_PPR3, 3a и 3b; NL_v2_PPR14, 14a и 14b; NL_v2_PPR 40, 40a и 40b; NL_v2_PPR12, 12a и 12b; NL_v2_PPR13, 13a и 13b; NL_v2_PPR2, 2a и 2b; NL_v2_PPR38, 38a и 38b; NL_v2_PPR37, 37a и 37b, NL_v2_PPR39, 39a и 39b; NL_v2_PPR56, 56a и 56b; и NL_v2_PPR68, 68a и 68b, SEQ ID NOS: 56 и 299-324). Для анализа с помощью RPB-ELISA используют биотинилированные РНК зонды, содержащие последовательность-мишень (T_1, T_49, T_3, T_14, T_40, T_12, T_13, T_2, T_38, T_37, T_39, T_56 и T_68) и биотинилированный РНК зонд (РНК 51, SEQ ID NO: 247), содержащий последовательность вне мишени (T_51, SEQ ID NO: 143), и анализируют активность связывания соответствующих белков PPR с мишенью (мишень) и вне мишени (вне мишени) с помощью RPB-ELISA.
Результаты для каждой последовательности-мишени показаны на фиг. 12A. Средние значения для каждого из белков с 18, 15 и 12 мотивами представлены в виде прямоугольной диаграммы, показанной на фиг. 12B. Более высокое количество мотивов обеспечивает более высокую силу связывания, и было определено, что белки с 18 мотивами делают возможным более стабильное получение белков с более высокой силой связывания по сравнению с белками с 18 и 15 мотивами.
[Пример 5: Искусственный контроль сплайсинга с использованием белков PPR]
Чтобы продемонстрировать, что белки PPR могут связываться с молекулой РНК-мишени в клетках и делать возможной желаемую манипуляцию с РНК, проводят эксперимент с использованием репортера сплайсинга (фиг. 13A). Репортер сплайсинга (RG6) имеет генетическую структуру, включающую экзон 1, интрон 1, экзон 2, интрон 2, экзон 3 и т.д. (Orengo et al., 2006 NAR). В интрон 1, экзон 2 и интрон 2, интрон 4 и интрон 5 cTNT курицы вставляют искусственно созданную альтернативная последовательность экзона. Этот репортер имел две формы сплайсинга, и соотношение количеств мРНК с и без проскока экзона 2 составляет примерно 1:1. Гены RFP и GFP кодированы в экзоне 3. В связи с этим, рамка считывания изменяется в зависимости от присутствия или отсутствия экзона 2, так что RFP экспрессируется с мРНК, в которой экзон 2 пропущен, а GFP экспрессируется с мРНК, в которой экзон 2 не пропущен. Известно, что количество форм сплайсинга этого репортера контролируется факторами сплайсинга, которые связываются с областями интрона 1, экзона 2 и интрона 2 (Orengo et al., 2006 NAR). Таким образом, отбирают 18 нуклеотидных последовательностей из областей интрона 1, экзона 2 и интрона 2, и исследуют, может ли форма сплайсинга репортера RG-6 быть изменена белками PPR, которые связываются с этими последовательностями.
Семь видов последовательностей-мишеней (Т77-Т83, SEQ ID NOS: 325-330) выбирают из репортера RG6. Гены белка PPR конструируют с мотивами как v1, так и v2 (v1_PPRsp1-6 и v2_PPRsp1-6, SEQ ID NOS: 331-342). Гены белка клонируют в pcDNA3.1, так чтобы экспрессировались белки, слитые с сигналом ядерной локализации на N-конце и с последовательностью метки эпитопа FLAG на C-конце (NLS_v1PPRsp1-6 и NLS_v2PPRsp1-6, SEQ ID NOS: 343-354). pcDNA3.1 имеет промотор CMV и сигнал poly-A SV40 (терминатор), между которыми вставлен ген белка PPR.
Клетки HEK293T инокулируют с плотностью 1 × 106 клеток в 10-сантиметровую чашку, содержащую 9 мл DMEM и 1 мл FBS, и культивируют в среде 37°C и 5% CO2 в течение 2 дней, и затем клетки собирают. Собранные клетки инокулируют на покрытый PLL 96-луночный планшет с плотностью 4 × 104 клеток/лунку и культивируют в среде 37°C и 5% CO2 в течение 1 дня. Готовят смесь из 100 нг ДНК плазмидной экспрессии PPR, 100 нг RG-6, 0,6 мкл Fugene (зарегистрированная торговая марка)-HD (Promega, E2311) и 200 мкл Opti-MEM, весь ее объем добавляют в лунки и культивирование проводят в среде 37°C и 5% CO2 в течение 2 дней. В качестве контроля также готовят образец, не содержащий PPR ДНК плазмиды экспрессии. После культивирования получают флуоресцентные изображения GFP и RFP каждой лунки с использованием флуоресцентного микроскопа DMi8 (Leica). Что касается условий визуализации, время экспозиции и усиление, при которых интенсивности GFP и RFP становятся практически одинаковыми, сначала определяют с использованием образца, содержащего только плазмиду RG-6, и флуоресцентные изображения образцов получают в тех же условиях.
После получения изображения, суммарную РНК экстрагируют с использованием набора Maxwell (зарегистрированная торговая марка) RSC simplyRNA Cells Kit. В 0,2 мл пробирку добавляют 500 нг экстрагированной общей РНК, 0,5 мкл 100 мкМ праймера dT20 и 0,5 мкл 10 мМ dNTP, оставляют при 65°C на 5 минут и немедленно охлаждают на льду. Туда добавляют 2 мкл 5x RT-буфера (Invitrogen, 18080-051), 0,5 мкл 0,1 M DTT, 0,5 мкл 40 Ед/мкл RNaseOUT (Invitrogen, 18080-051) и 0,5 мкл 200 единиц/мкл SupperScript III. (Invitrogen, 18080-051), и реакцию проводят в термоциклере при 50°C в течение 50 минут, затем при 85°C в течение 5 минут и охлаждают до 16°C. Образец с обратной транскрипцией разводят 10-кратно стерильной водой. В 0,2 мл пробирку добавляют 2 мкл реакционной смеси, 10 мкл 5х буфера GXL (TAKARA, R050A), 4 мкл 2,5 мМ dNTP, 1,5 мкл 10 мкМ праймера RT-Fw (5'-CAAAGTGGAGGACCCAGTACC-3', SEQ ID NO: 355), 1,5 мкл 10 мкМ праймера RT-Rv (5'-GCGCATGAACTCCTTGATGAC-3', SEQ ID NO: 356), 1 мкл GXL (TAKARA, R050A) и 31,5 мкл стерильной воды, реакцию проводят в термоциклере при 98°C в течение 2 минут, затем проводят 35 циклов при 98°C в течение 10 секунд, 58°C в течение 15 секунд и 68°C в течение 5 секунд, и затем реакционную смесь охлаждают до 12°C. Реакционную смесь разбавляют 10-кратно и подвергают электрофорезу с MultiNA (SHIMADZU, MCE202). Полосу длиной примерно 114 п.н. и полосу примерно 142 п.н. рассматривают как полосу РНК с пропущенным экзоном и полосу РНК без проскока, соответственно, и рассчитывают интенсивности полос образцов. Величина, полученная делением интенсивности полосы 114 п.о. на сумму интенсивности полосы 114 п.о. и интенсивности полосы 142 п.о., определяют как коэффициент сплайсинга.
Результаты представлены на фиг. 13B и 13C. Определено, что коэффициент сплайсинга составляет 0,48 при введении только репортера RG6, и является аналогичным при введении PPRsp4, но значительно изменяется при введении других PPR. По сравнению с v1 и v2, v2 обеспечивает большее изменение, за исключением PPRsp4. Эти соотношения сплайсинга также соответствуют отношениям экспрессии RFP и GFP, показанным на фиг. 13B. Эти результаты подтверждают, что белки PPR могут применяться для изменения проскока экзонов, и показывают, что мотив v2 может использоваться для изменения сплайсинга еще более эффективно.
[Пример 6: Регулирование агрегации белка PPR]
Белок PPR с использованием мотива V2 (SEQ ID NO: 457 для нуклеотидной последовательности и SEQ ID NO: 458 для аминокислотной последовательности) и белок PPR с использованием мотива v3.2 (SEQ ID NO: 459 для нуклеотидной последовательности и SEQ ID NO: 460 для аминокислотной последовательности) получают в системе экспрессии E. coli, соответственно, очищают и разделяют гель-фильтрационной хроматографией.
(Экспрессия и очистка белков)
Штамм Розетта E.coli трансформируют с плазмидой pE-SUMOpro Kan, содержащей последовательность ДНК, кодирующую целевой белок PPR, и культивируют при 37°С, затем температуру понижают до 20°C, когда OD600 достигает 0,6, и добавляют IPTG в конечной концентрации 0,5 мМ, чтобы целевой PPR экспрессировался в клетках E. coli как слитый с SUMO белок. Клетки культивируют в течение ночи, затем собирают центрифугированием и ресуспендируют в буфере для лизиса (50 мМ Tris-HCl, pH 8,0, 500 мМ NaCl). Клетки E.coli разрушают ультразвуком и центрифугируют при 17000g в течение 30 минут, после чего фракцию супернатанта наносят на колонку Ni-Agarose, колонку промывают буфером для лизиса, содержащим 20 мМ имидазола, и затем SUMO-слитый целевой белок PPR элюируют буфером для лизиса, содержащим 400 мМ имидазола. После элюирования белок SUMO отщепляют от целевого белка PPR с помощью Ulp1, и в то же время раствор белка меняют на ионообменный буфер (50 мМ Tris-HCl, pH 8,0, 200 мМ NaCl) путем диализа. Затем проводят катионообменную хроматографию с использованием колонки SP. После нанесения на колонку, белки элюируют постепенно увеличивающейся концентрацией NaCl от 200 мМ до 1 М. Фракцию, содержащую целевой белок PPR, подвергают окончательной очистке гель-фильтрационной хроматографией с использованием колонки Superdex 200. Целевой белок PPR, элюированный из ионообменной колонки, наносят на колонку для гель-фильтрации, уравновешенную буфером для гель-фильтрации (25 мМ HEPES, pH 7,5, 200 мМ NaCl, 0,5 мМ трис(2-карбоксиэтил)фосфина (TCEP)). Наконец, фракцию, содержащую целевой белок PPR, концентрируют, замораживают в жидком азоте и хранят при -80°C до использования для следующего анализа.
(Гель-фильтрационная хроматография)
Очищенный рекомбинантный белок PPR получают в концентрации 1 мг/мл. Для гель-фильтрационной хроматографии, используют увеличение Superdex 200 10/300 GL (GE Healthcare). На колонку для гель-фильтрации, уравновешенную 25 мМ HEPES pH 7,5, 200 мМ NaCl, 0,5 мМ трис(2-карбоксиэтил)фосфина (TCEP), наносят приготовленный белок, и абсорбцию раствора, элюированного из колонки для гель-фильтрации, измеряют при 280 нм для анализа свойств белка.
(Полученные результаты)
Результаты показаны на фиг. 14. Меньший объем фракции элюирования (объем элюирования) означает больший размер молекулы. Белок с использованием v2 элюируют во фракциях элюирования 8-10 мл, тогда как пик белка с использованием v3.2 наблюдают во фракциях элюирования 12-14 мл. Этот результат предполагает возможность того, что белок, использующий v2, агрегируется из-за его большего размера, и агрегация улучшена в белке, использующем v3.2.
| название | год | авторы | номер документа |
|---|---|---|---|
| ПЕСТИЦИДНЫЕ КОМПОЗИЦИИ И ОТНОСЯЩИЕСЯ К НИМ СПОСОБЫ | 2013 |
|
RU2662672C2 |
| НОВЫЙ АКТИВАТОР ТРАНСКРИПЦИИ | 2019 |
|
RU2800921C2 |
| КОНСТРУКЦИИ ГОМОДИМЕРНЫХ БЕЛКОВ | 2011 |
|
RU2624041C2 |
| УЛУЧШЕННЫЕ N-ТЕРМИНАЛЬНЫЕ КЭППИНГ МОДУЛИ ДЛЯ СКОНСТРУИРОВАННЫХ БЕЛКОВ С АНКИРИНОВЫМ ПОВТОРОМ | 2011 |
|
RU2636552C2 |
| ВАКЦИНЫ ПРОТИВ HPV | 2012 |
|
RU2644201C2 |
| МУТАНТЫ ИНДУЦИРУЕМОЙ АКТИВАЦИЕЙ ЦИТИДИНДЕАМИНАЗЫ (AID) И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2010 |
|
RU2537264C2 |
| НЕЙРОТОКСИНЫ, ПРОЯВЛЯЮЩИЕ УКОРОЧЕННУЮ БИОЛОГИЧЕСКУЮ АКТИВНОСТЬ | 2012 |
|
RU2646110C2 |
| ВАРИАНТЫ B4GALT1 И ИХ ПРИМЕНЕНИЕ | 2018 |
|
RU2805557C2 |
| ГЛИКОЗИЛИРОВАННЫЕ КОНЪЮГАТЫ МОЛЕКУЛ С ПОВТОРЯЮЩИМСЯ МОТИВОМ | 2010 |
|
RU2574201C2 |
| СВЯЗЫВАЮЩИЙ RGMA БЕЛОК И ЕГО ИСПОЛЬЗОВАНИЕ | 2016 |
|
RU2705304C2 |
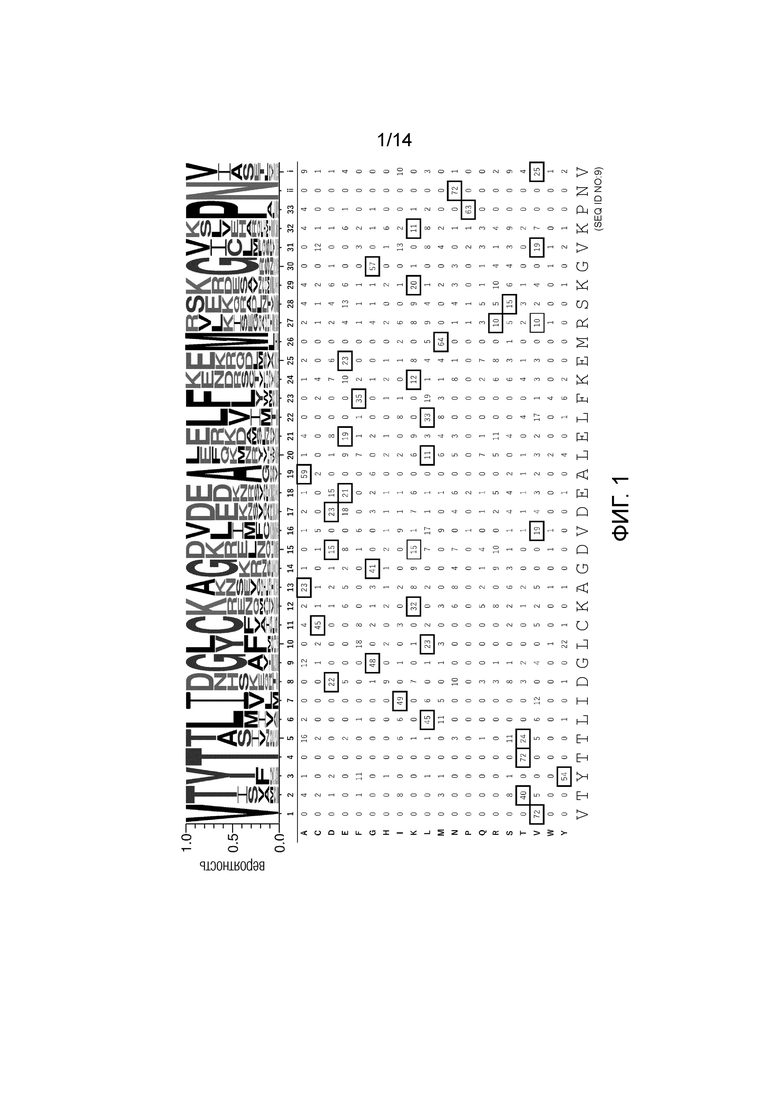
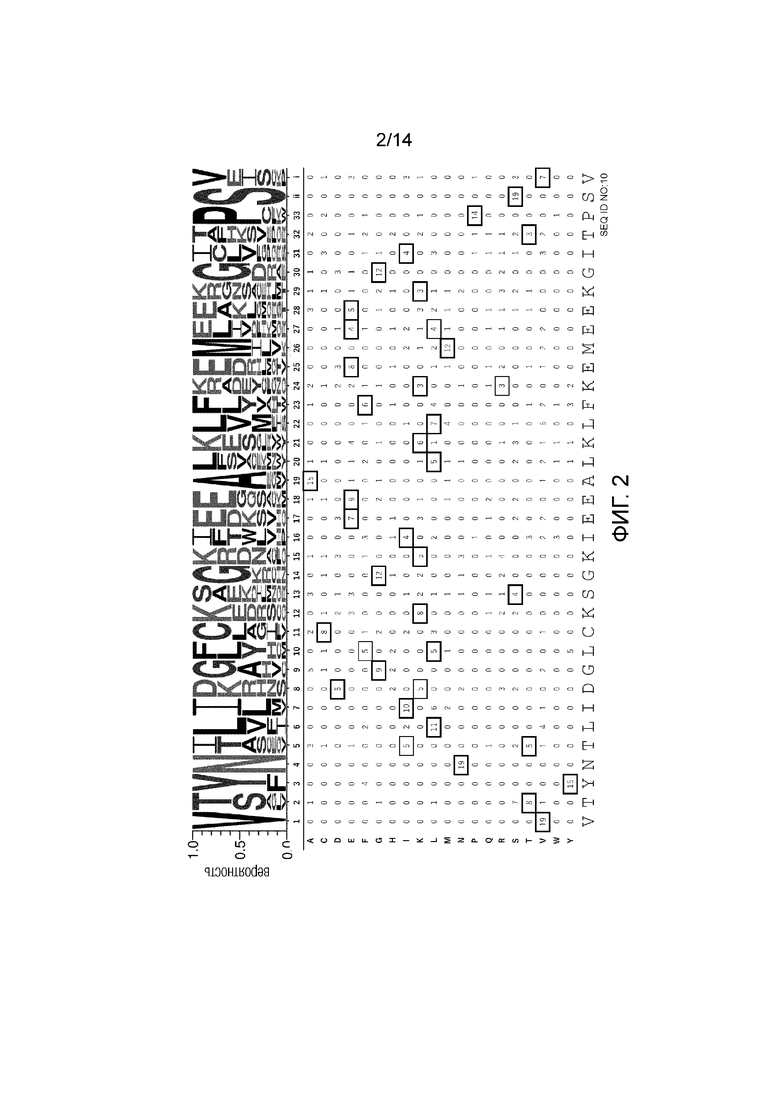
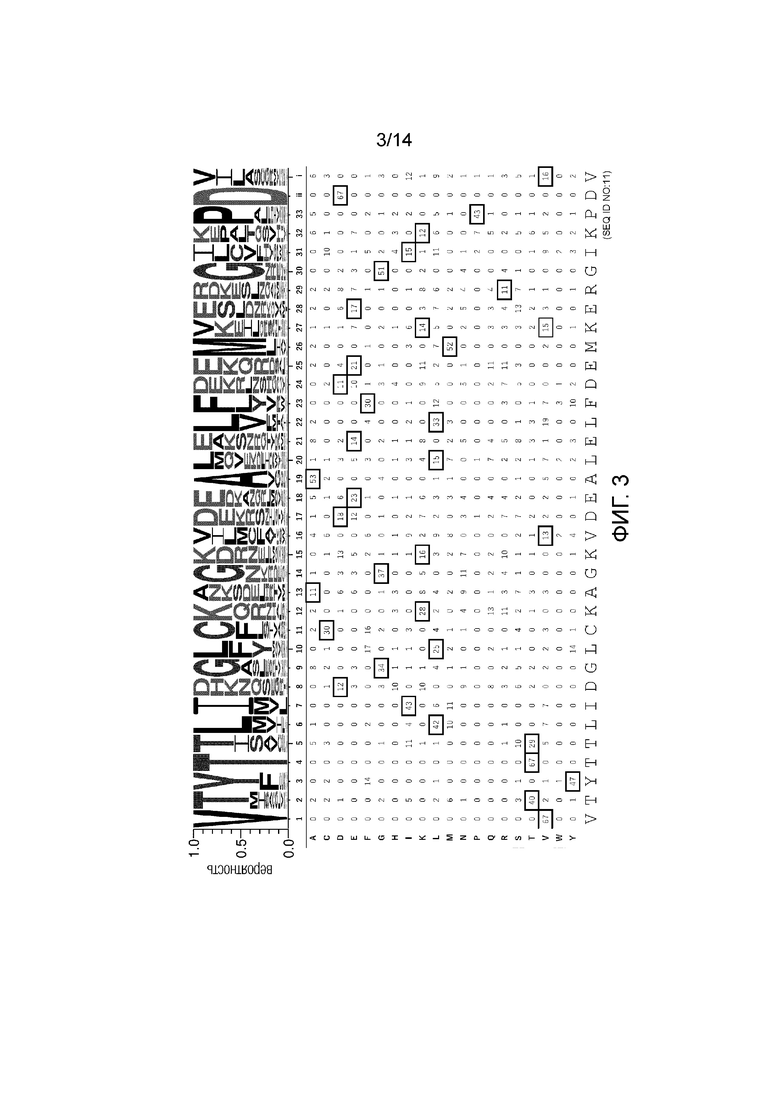
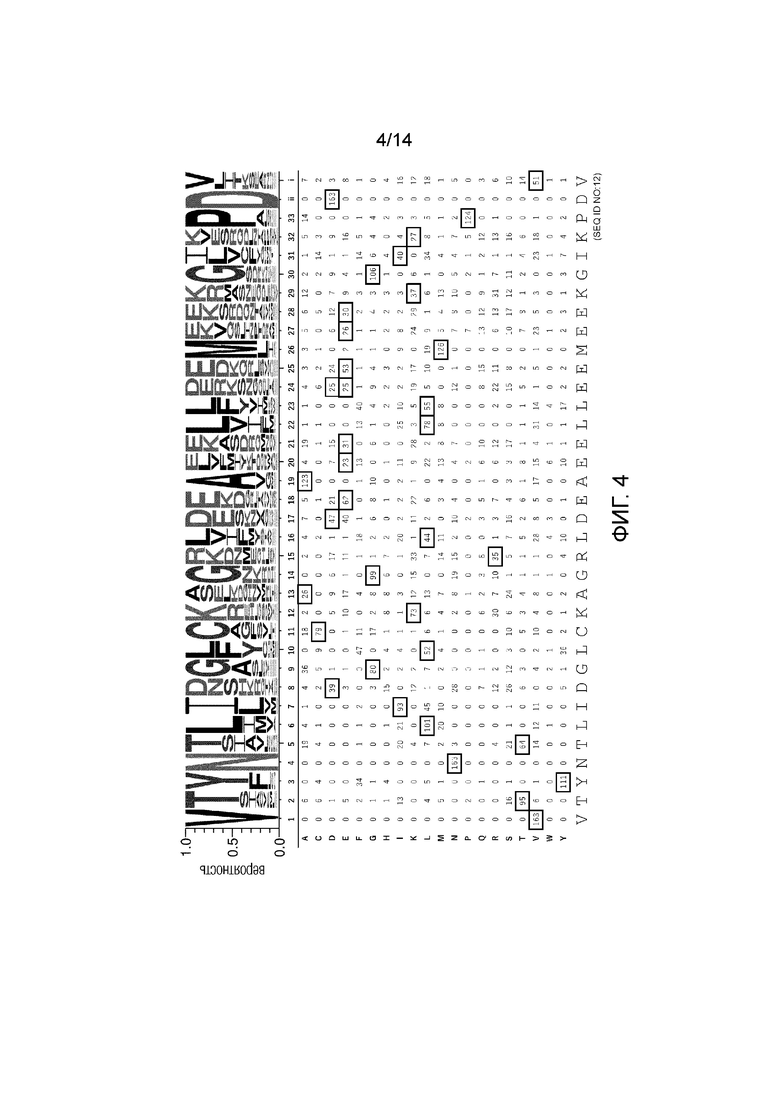
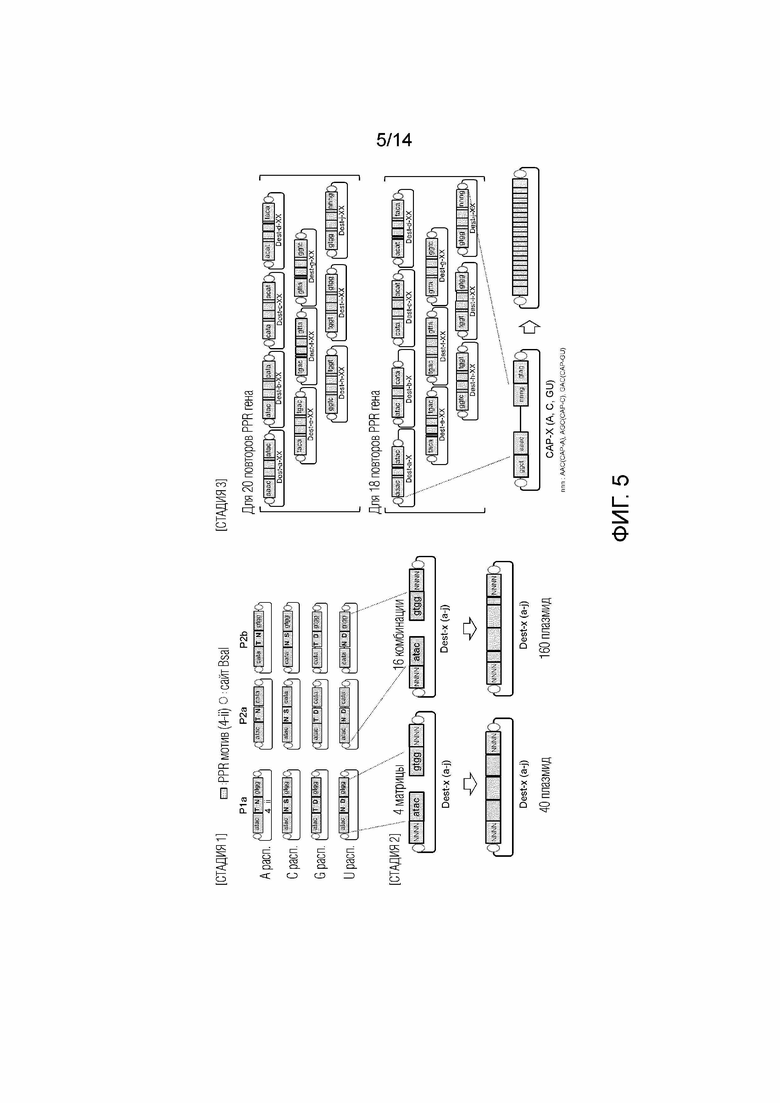
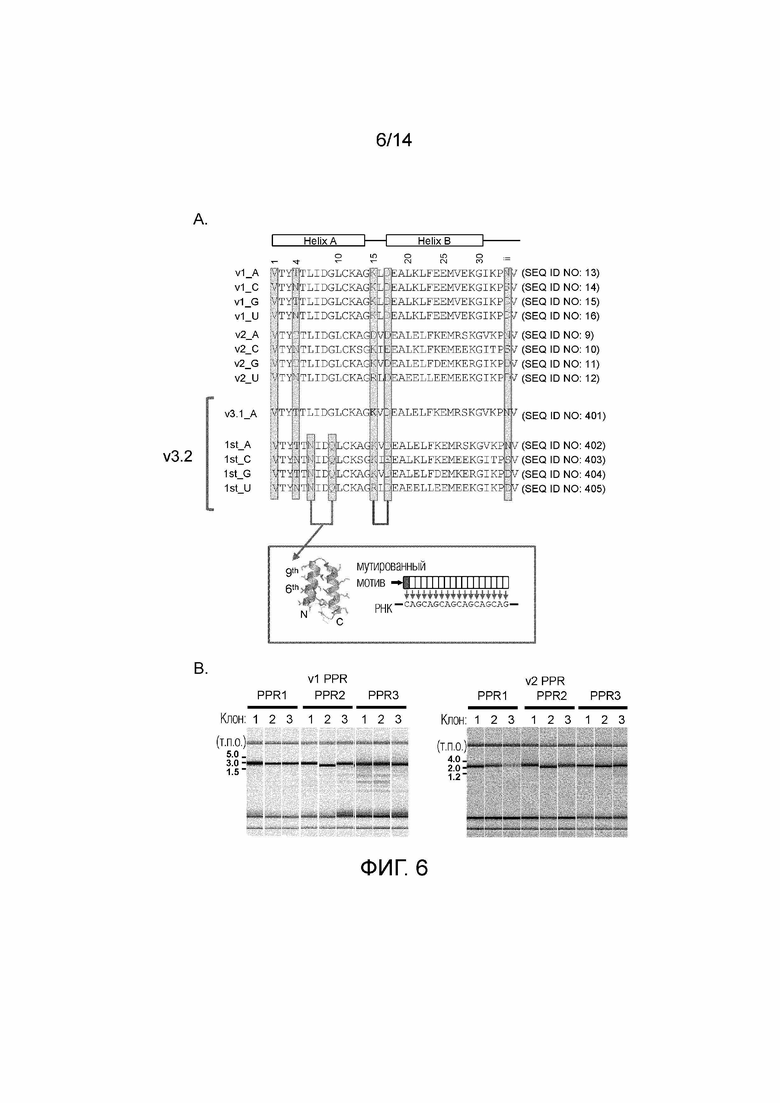
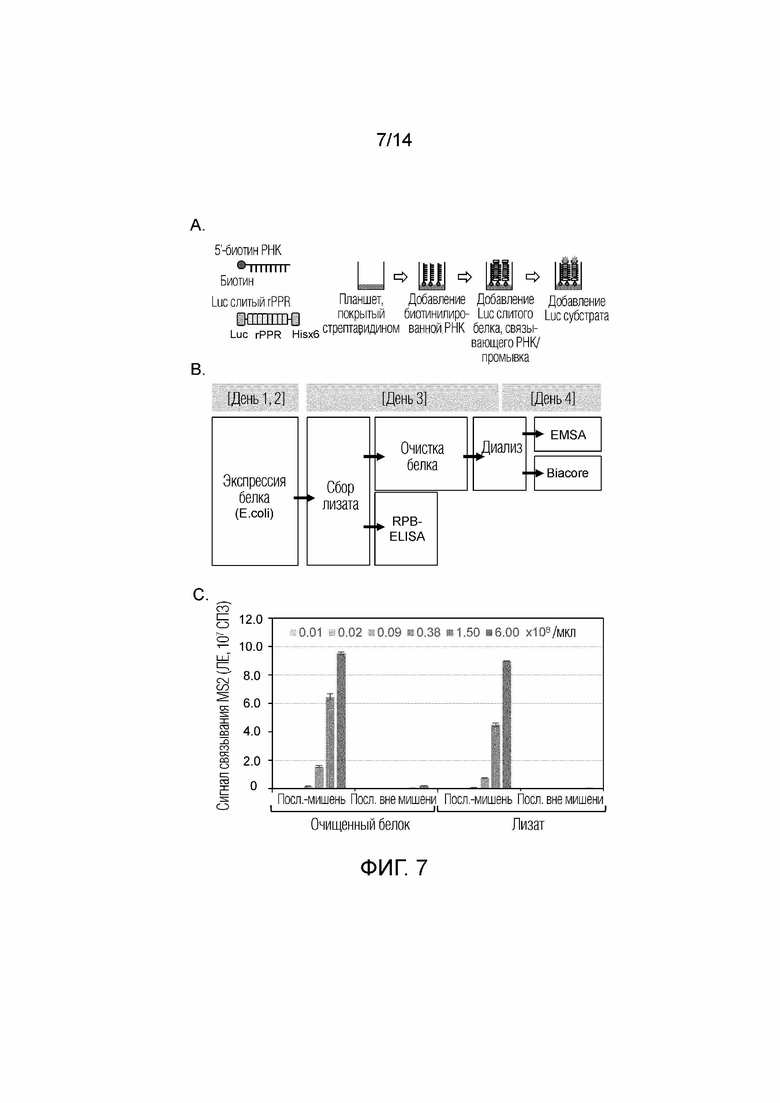
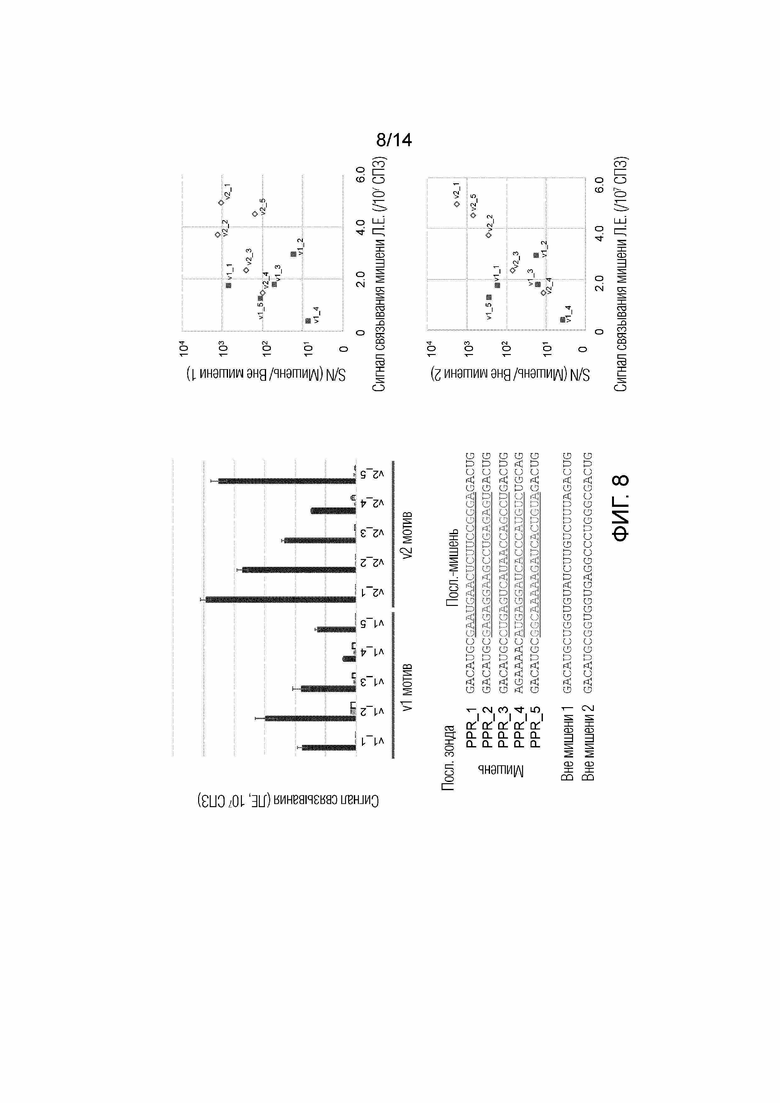
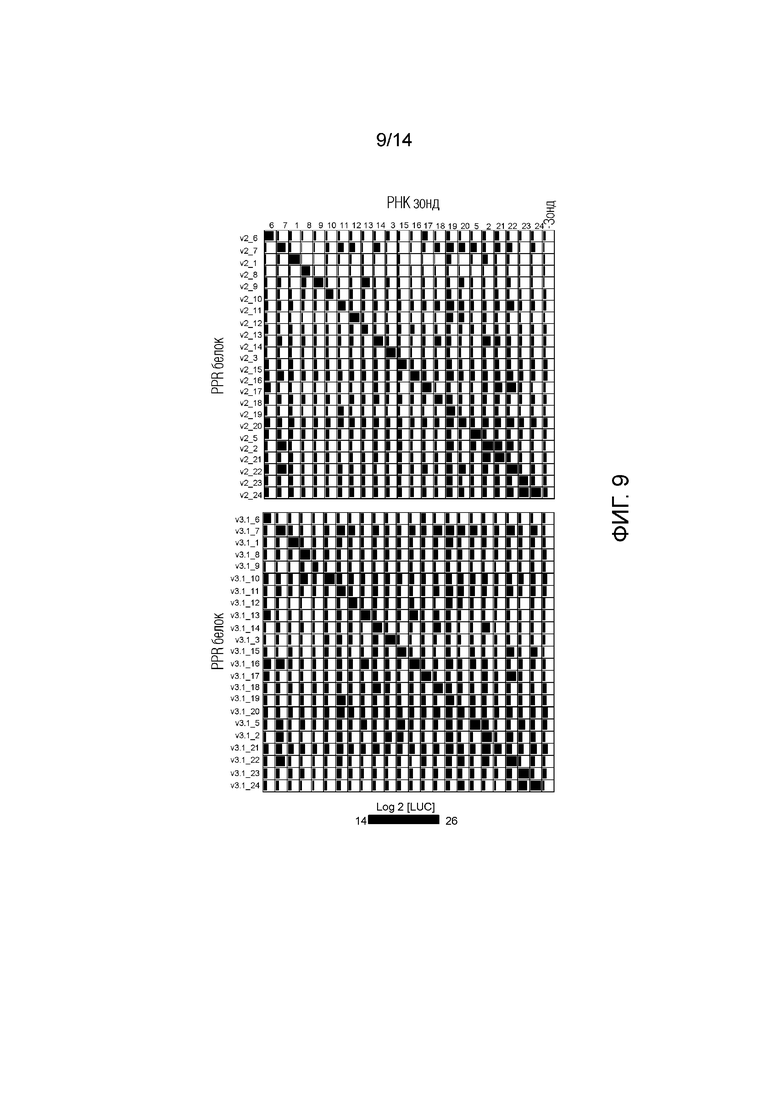
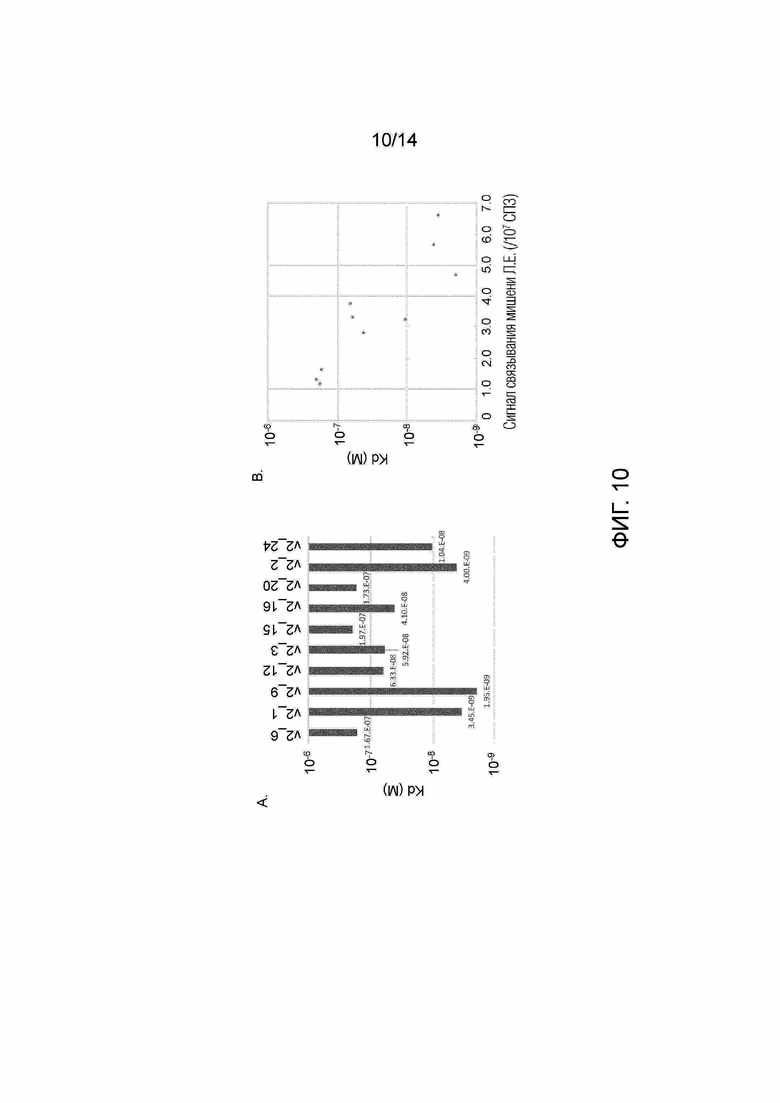
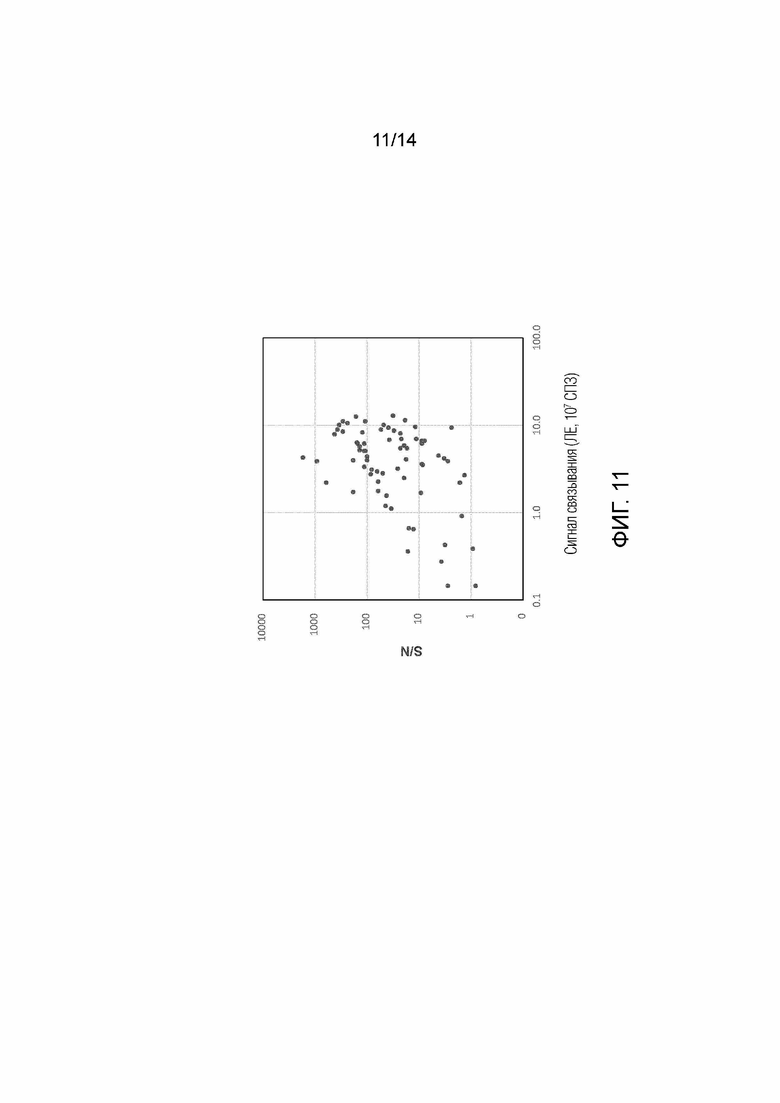


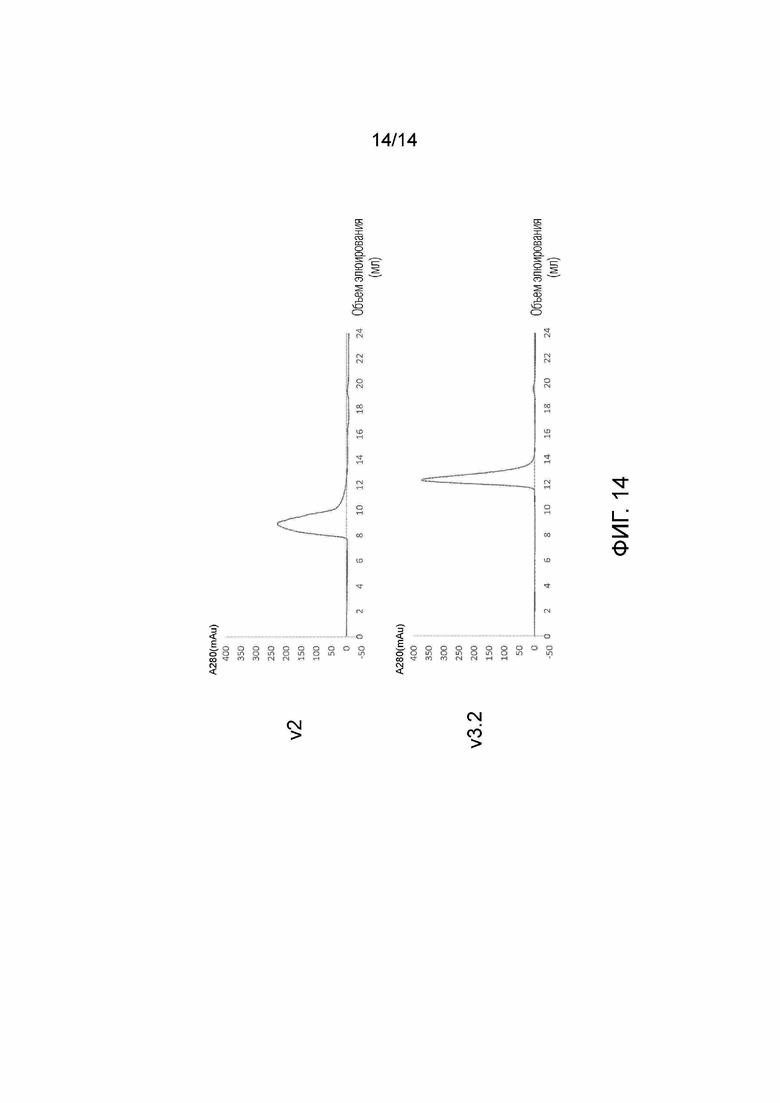
Изобретение относится к области биотехнологии, конкретно к белкам с пентатрикопептидными повторами (PPR), и может быть использовано для контроля сплайсинга РНК, выявления РНК, манипулирования РНК и получения организма, не являющегося человеком. Предложены рекомбинантные белки и полипептиды с мотивами PPR, связывающими аденин, гуанин, цитозин и урацил. Изобретение обеспечивает получение белков и полипептидов, специфически связывающихся с РНК-мишенью в клетках с высокой эффективностью. 12 н. и 1 з.п. ф-лы, 14 ил., 10 табл., 6 пр.
1. Белок, специфически связывающийся с молекулой РНК-мишенью в клетках, где указанный белок содержит по меньшей мере два полипептида, каждый из которых является любым из следующих:
(A-1) полипептид, состоящий из последовательности SEQ ID NO: 9, или полипептид, состоящий из последовательности SEQ ID NO: 9, имеющий замену, выбранную из группы, состоящей из аминокислотной замены в положении 10 тирозином, аминокислотной замены в положении 15 лизином, аминокислотной замены в положении 16 лейцином, аминокислотной замены в положении 17 глутаминовой кислотой, аминокислотной замены в положении 18 аспарагиновой кислотой и аминокислотной замены в положении 28 глутаминовой кислотой; или
полипептид, состоящий из последовательности SEQ ID NO: 401, или полипептид, состоящий из последовательности SEQ ID NO: 401, имеющей замену, выбранную из группы, состоящей из аминокислотной замены в положении 10 тирозином, аминокислотной замены в положении 16 лейцином, аминокислотной замены в положении 17 глутаминовой кислотой, аминокислотной замены в положении 18 аспарагиновой кислотой и аминокислотной замены в положении 28 глутаминовой кислотой;
(A-2) полипептид, состоящий из последовательности SEQ ID NO: 9 или 401, имеющей замену, делецию или добавление от 1 до 4 аминокислот, отличных от аминокислот в положениях 1, 2, 3, 4, 6, 7, 9, 11, 12, 14, 19, 26, 30, 33 и 34, и обладающий свойством связывания аденина, где полипептид представлен Формулой 1, приведенной ниже;
(A-3) полипептид, имеющий идентичность последовательности по меньшей мере 90% с последовательностью SEQ ID NO: 9 или 401, при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 7, 9, 11, 12, 14, 19, 26, 30, 33 и 34 идентичны, и обладающий свойством связывания аденина, где полипептид представлен Формулой 1, приведенной ниже;
(C-1) полипептид, состоящий из последовательности SEQ ID NO: 10, или полипептид, состоящий из последовательности SEQ ID NO: 10, имеющей аминокислотную замену, выбранную из группы, состоящей из аминокислотной замены в положении 2 серином, аминокислотной замены в положении 5 изолейцином, аминокислотной замены в положении 7 лейцином, аминокислотной замены в положении 8 лизином, аминокислотной замены в положении 10 фенилаланином или тирозином, аминокислотной замены в положении 15 аргинином, аминокислотной замены в положении 22 валином, аминокислотной замены в положении 24 аргинином, аминокислотной замены в положении 27 лейцином и аминокислотной замены в положении 29 аргинином;
(C-2) полипептид, состоящий из последовательности SEQ ID NO: 10, имеющей замену, делецию или добавление от 1 до 4 аминокислот, отличных от аминокислот в положениях 1, 3, 4, 14, 18, 19, 26, 30, 33 и 34, и обладающий свойством связывания цитозина, где полипептид представлен Формулой 1, приведенной ниже;
(C-3) полипептид, имеющий идентичность последовательности по меньшей мере 90% с последовательностью SEQ ID NO: 10, при условии, что аминокислоты в положениях 1, 3, 4, 14, 18, 19, 26, 30, 33 и 34 идентичны, и обладающий свойством связывания цитозина, где полипептид представлен Формулой 1, приведенной ниже;
(G-1) полипептид, состоящий из последовательности SEQ ID NO: 11, или полипептид, состоящий из последовательности SEQ ID NO: 11, имеющий замену, выбранную из группы, состоящей из аминокислотной замены в положении 10 фенилаланином, аминокислотной замены в положении 15 аспарагиновой кислотой, аминокислотной замены в положении 27 валином, аминокислотной замены в положении 28 серином и аминокислотной замены в положении 35 изолейцином;
(G-2) полипептид, состоящий из последовательности SEQ ID NO: 11, имеющей замену, делецию или добавление от 1 до 4 аминокислот, отличных от аминокислот в положениях 1, 2, 3, 4, 6, 7, 9, 14, 18, 19, 26, 30, 33 и 34, и обладающий свойством связывания гуанина, где полипептид представлен Формулой 1, приведенной ниже;
(G-3) полипептид, имеющий идентичность последовательности по меньшей мере 90% с последовательностью SEQ ID NO: 11, при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 7, 9, 14, 18, 19, 26, 30, 33 и 34 идентичны, и обладающий свойством связывания гуанина, где полипептид представлен Формулой 1, приведенной ниже;
(U-1) полипептид, состоящий из последовательности SEQ ID NO: 12, или полипептид, состоящий из последовательности SEQ ID NO: 12, имеющий замену, выбранную из группы, состоящей из аминокислотной замены в положении 10 фенилаланином, аминокислотной замены в положении 13 серином, аминокислотной замены в положении 15 лизином, аминокислотной замены в положении 17 глутаминовой кислотой, аминокислотной замены в положении 20 лейцином, аминокислотной замены в положении 21 лизином, аминокислотной замены в положении 23 фенилаланином, аминокислотной замены в положении 24 аспарагиновой кислотой, аминокислотной замены в положении 27 лизином, аминокислотной замены в положении 28 лизином, аминокислотной замены в положении 29 аргинином и аминокислотной замены в положении 31 лейцином;
(U-2) полипептид, состоящий из последовательности SEQ ID NO: 12, имеющей замену, делецию или добавление от 1 до 4 аминокислот, кроме аминокислот в положениях 1, 2, 3, 4, 6, 11, 12, 14, 19, 26, 30, 33 и 34, и обладающий свойством связывания урацила, где полипептид представлен Формулой 1, приведенной ниже; и
(U-3) полипептид, имеющий идентичность последовательности по меньшей мере 90% с последовательностью SEQ ID NO: 12, при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 11, 12, 14, 19, 26, 30, 33 и 34 идентичны, и обладающий свойством связывания урацила, где полипептид представлен Формулой 1, приведенной ниже,
(Спираль А)-Х-(Спираль В)-L, Формула 1
в Формуле:
Спираль A является группой длиной 12 аминокислот, способной образовывать структуру α-спирали, и представлена формулой 2
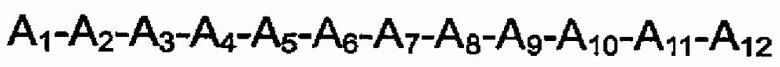 , Формула 2
, Формула 2
где в формуле 2 A1-A12 независимо являются аминокислотой;
X не существует или является группой длиной от 1 до 9 аминокислот;
Спираль B является группой длиной от 11 до 13 аминокислот, способной образовывать структуру α-спирали; и
L является группой длиной от 2 до 7 аминокислот, представленной формулой 3
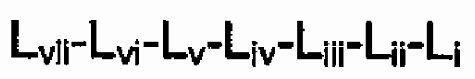 , Формула 3
, Формула 3
где в формуле 3 аминокислоты пронумерованы «i» (-1), «ii» (-2) и так далее со стороны С-конца,
при условии, что Liii-Lvii могут не существовать.
2. Белок по п. 1, где по меньшей мере два полипептида выбирают из любого из полипептидов, состоящих из последовательности SEQ ID NO: 9-12 или 401.
3. Применение полипептида для получения белка c пентатрикопептидными повторами (PPR), РНК-мишень которого имеет длину 15 оснований или более, где полипептид выбирают из следующих:
(A-1) полипептид, состоящий из последовательности SEQ ID NO: 9, или полипептид, состоящий из последовательности SEQ ID NO: 9, имеющий замену, выбранную из группы, состоящей из аминокислотной замены в положении 10 тирозином, аминокислотной замены в положении 15 лизином, аминокислотной замены в положении 16 лейцином, аминокислотной замены в положении 17 глутаминовой кислотой, аминокислотной замены в положении 18 аспарагиновой кислотой и аминокислотной замены в положении 28 глутаминовой кислотой; или
полипептид, состоящий из последовательности SEQ ID NO: 401, или полипептид, состоящий из последовательности SEQ ID NO: 401, имеющей замену, выбранную из группы, состоящей из аминокислотной замены в положении 10 тирозином, аминокислотной замены в положении 16 лейцином, аминокислотной замены в положении 17 глутаминовой кислотой, аминокислотной замены в положении 18 аспарагиновой кислотой и аминокислотной замены в положении 28 глутаминовой кислотой;
(A-2) полипептид, состоящий из последовательности SEQ ID NO: 9 или 401, имеющей замену, делецию или добавление от 1 до 4 аминокислот, отличных от аминокислот в положениях 1, 2, 3, 4, 6, 7, 9, 11, 12, 14, 19, 26, 30, 33 и 34, и обладающий свойством связывания аденина, где полипептид представлен Формулой 1, приведенной ниже;
(A-3) полипептид, имеющий идентичность последовательности по меньшей мере 90% с последовательностью SEQ ID NO: 9 или 401, при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 7, 9, 11, 12, 14, 19, 26, 30, 33 и 34 идентичны, и обладающий свойством связывания аденина, где полипептид представлен Формулой 1, приведенной ниже;
(C-1) полипептид, состоящий из последовательности SEQ ID NO: 10, или полипептид, состоящий из последовательности SEQ ID NO: 10, имеющей аминокислотную замену, выбранную из группы, состоящей из аминокислотной замены в положении 2 серином, аминокислотной замены в положении 5 изолейцином, аминокислотной замены в положении 7 лейцином, аминокислотной замены в положении 8 лизином, аминокислотной замены в положении 10 фенилаланином или тирозином, аминокислотной замены в положении 15 аргинином, аминокислотной замены в положении 22 валином, аминокислотной замены в положении 24 аргинином, аминокислотной замены в положении 27 лейцином и аминокислотной замены в положении 29 аргинином;
(C-2) полипептид, состоящий из последовательности SEQ ID NO: 10, имеющей замену, делецию или добавление от 1 до 4 аминокислот, отличных от аминокислот в положениях 1, 3, 4, 14, 18, 19, 26, 30, 33 и 34, и обладающий свойством связывания цитозина, где полипептид представлен Формулой 1, приведенной ниже;
(C-3) полипептид, имеющий идентичность последовательности по меньшей мере 90% с последовательностью SEQ ID NO: 10, при условии, что аминокислоты в положениях 1, 3, 4, 14, 18, 19, 26, 30, 33 и 34 идентичны, и обладающий свойством связывания цитозина, где полипептид представлен Формулой 1, приведенной ниже;
(G-1) полипептид, состоящий из последовательности SEQ ID NO: 11, или полипептид, состоящий из последовательности SEQ ID NO: 11, имеющий замену, выбранную из группы, состоящей из аминокислотной замены в положении 10 фенилаланином, аминокислотной замены в положении 15 аспарагиновой кислотой, аминокислотной замены в положении 27 валином, аминокислотной замены в положении 28 серином и аминокислотной замены в положении 35 изолейцином;
(G-2) полипептид, состоящий из последовательности SEQ ID NO: 11, имеющей замену, делецию или добавление от 1 до 4 аминокислот, отличных от аминокислот в положениях 1, 2, 3, 4, 6, 7, 9, 14, 18, 19, 26, 30, 33 и 34, и обладающий свойством связывания гуанина, где полипептид представлен Формулой 1, приведенной ниже;
(G-3) полипептид, имеющий идентичность последовательности по меньшей мере 90% с последовательностью SEQ ID NO: 11, при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 7, 9, 14, 18, 19, 26, 30, 33 и 34 идентичны, и обладающий свойством связывания гуанина, где полипептид представлен Формулой 1, приведенной ниже;
(U-1) полипептид, состоящий из последовательности SEQ ID NO: 12, или полипептид, состоящий из последовательности SEQ ID NO: 12, имеющий замену, выбранную из группы, состоящей из аминокислотной замены в положении 10 фенилаланином, аминокислотной замены в положении 13 серином, аминокислотной замены в положении 15 лизином, аминокислотной замены в положении 17 глутаминовой кислотой, аминокислотной замены в положении 20 лейцином, аминокислотной замены в положении 21 лизином, аминокислотной замены в положении 23 фенилаланином, аминокислотной замены в положении 24 аспарагиновой кислотой, аминокислотной замены в положении 27 лизином, аминокислотной замены в положении 28 лизином, аминокислотной замены в положении 29 аргинином и аминокислотной замены в положении 31 лейцином;
(U-2) полипептид, состоящий из последовательности SEQ ID NO: 12, имеющей замену, делецию или добавление от 1 до 4 аминокислот, кроме аминокислот в положениях 1, 2, 3, 4, 6, 11, 12, 14, 19, 26, 30, 33 и 34, и обладающий свойством связывания урацила, где полипептид представлен Формулой 1, приведенной ниже; и
(U-3) полипептид, имеющий идентичность последовательности по меньшей мере 90% с последовательностью SEQ ID NO: 12, при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 11, 12, 14, 19, 26, 30, 33 и 34 идентичны, и обладающий свойством связывания урацила, где полипептид представлен Формулой 1, приведенной ниже,
(Спираль А)-Х-(Спираль В)-L, Формула 1
в Формуле:
Спираль A является группой длиной 12 аминокислот, способной образовывать структуру α-спирали, и представлена формулой 2
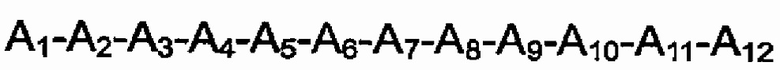 , Формула 2
, Формула 2
где в формуле 2 A1-A12 независимо являются аминокислотой;
X не существует или является группой длиной от 1 до 9 аминокислот;
Спираль B является группой длиной от 11 до 13 аминокислот, способной образовывать структуру α-спирали; и
L является группой длиной от 2 до 7 аминокислот, представленной формулой 3
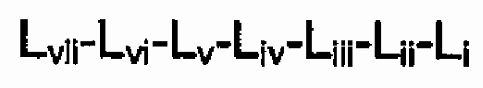 , Формула 3
, Формула 3
где в формуле 3 аминокислоты пронумерованы «i» (-1), «ii» (-2) и так далее со стороны С-конца,
при условии, что Liii-Lvii могут не существовать.
4. Белок, специфически связывающийся с молекулой РНК-мишенью в клетках, где указанный белок содержит полипептид, состоящий из n мотивов (PPR), отличающийся тем, что:
мотивом PPR для аденина в нуклеотидной последовательности является один из следующих:
(A-1) полипептид, состоящий из последовательности SEQ ID NO: 9, или полипептид, состоящий из последовательности SEQ ID NO: 9, имеющий замену, выбранную из группы, состоящей из аминокислотной замены в положении 10 тирозином, аминокислотной замены в положении 15 лизином, аминокислотной замены в положении 16 лейцином, аминокислотной замены в положении 17 глутаминовой кислотой, аминокислотной замены в положении 18 аспарагиновой кислотой и аминокислотной замены в положении 28 глутаминовой кислотой; или полипептид, состоящий из последовательности SEQ ID NO: 401, или полипептид, состоящий из последовательности SEQ ID NO: 401, имеющей замену, выбранную из группы, состоящей из аминокислотной замены в положении 10 тирозином, аминокислотной замены в положении 16 лейцином, аминокислотной замены в положении 17 глутаминовой кислотой, аминокислотной замены в положении 18 аспарагиновой кислотой и аминокислотной замены в положении 28 глутаминовой кислотой;
(A-2) полипептид, состоящий из последовательности SEQ ID NO: 9 или 401, имеющей замену, делецию или добавление от 1 до 4 аминокислот, отличных от аминокислот в положениях 1, 2, 3, 4, 6, 7, 9, 11, 12, 14, 19, 26, 30, 33 и 34, и обладающий свойством связывания аденина, где полипептид представлен Формулой 1, приведенной ниже;
(A-3) полипептид, имеющий идентичность последовательности по меньшей мере 90% с последовательностью SEQ ID NO: 9 или 401, при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 7, 9, 11, 12, 14, 19, 26, 30, 33 и 34 идентичны, и обладающий свойством связывания аденина, где полипептид представлен Формулой 1, приведенной ниже;
мотивом PPR для цитозина в нуклеотидной последовательности является один из следующих:
(C-1) полипептид, состоящий из последовательности SEQ ID NO: 10, или полипептид, состоящий из последовательности SEQ ID NO: 10, имеющей аминокислотную замену, выбранную из группы, состоящей из аминокислотной замены в положении 2 серином, аминокислотной замены в положении 5 изолейцином, аминокислотной замены в положении 7 лейцином, аминокислотной замены в положении 8 лизином, аминокислотной замены в положении 10 фенилаланином или тирозином, аминокислотной замены в положении 15 аргинином, аминокислотной замены в положении 22 валином, аминокислотной замены в положении 24 аргинином, аминокислотной замены в положении 27 лейцином и аминокислотной замены в положении 29 аргинином;
(C-2) полипептид, состоящий из последовательности SEQ ID NO: 10, имеющей замену, делецию или добавление от 1 до 4 аминокислот, отличных от аминокислот в положениях 1, 3, 4, 14, 18, 19, 26, 30, 33 и 34, и обладающий свойством связывания цитозина, где полипептид представлен Формулой 1, приведенной ниже;
(C-3) полипептид, имеющий идентичность последовательности по меньшей мере 90% с последовательностью SEQ ID NO: 10, при условии, что аминокислоты в положениях 1, 3, 4, 14, 18, 19, 26, 30, 33 и 34 идентичны, и обладающий свойством связывания цитозина, где полипептид представлен Формулой 1, приведенной ниже;
мотивом PPR для гуанина в нуклеотидной последовательности является один из следующих:
(G-1) полипептид, состоящий из последовательности SEQ ID NO: 11, или полипептид, состоящий из последовательности SEQ ID NO: 11, имеющий замену, выбранную из группы, состоящей из аминокислотной замены в положении 10 фенилаланином, аминокислотной замены в положении 15 аспарагиновой кислотой, аминокислотной замены в положении 27 валином, аминокислотной замены в положении 28 серином и аминокислотной замены в положении 35 изолейцином;
(G-2) полипептид, состоящий из последовательности SEQ ID NO: 11, имеющей замену, делецию или добавление от 1 до 4 аминокислот, отличных от аминокислот в положениях 1, 2, 3, 4, 6, 7, 9, 14, 18, 19, 26, 30, 33 и 34, и обладающий свойством связывания гуанина, где полипептид представлен Формулой 1, приведенной ниже;
(G-3) полипептид, имеющий идентичность последовательности по меньшей мере 90% с последовательностью SEQ ID NO: 11, при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 7, 9, 14, 18, 19, 26, 30, 33 и 34 идентичны, и обладающий свойством связывания гуанина, где полипептид представлен Формулой 1, приведенной ниже; и
мотивом PPR для урацила в нуклеотидной последовательности является один из следующих:
(U-1) полипептид, состоящий из последовательности SEQ ID NO: 12, или полипептид, состоящий из последовательности SEQ ID NO: 12, имеющий замену, выбранную из группы, состоящей из аминокислотной замены в положении 10 фенилаланином, аминокислотной замены в положении 13 серином, аминокислотной замены в положении 15 лизином, аминокислотной замены в положении 17 глутаминовой кислотой, аминокислотной замены в положении 20 лейцином, аминокислотной замены в положении 21 лизином, аминокислотной замены в положении 23 фенилаланином, аминокислотной замены в положении 24 аспарагиновой кислотой, аминокислотной замены в положении 27 лизином, аминокислотной замены в положении 28 лизином, аминокислотной замены в положении 29 аргинином и аминокислотной замены в положении 31 лейцином;
(U-2) полипептид, состоящий из последовательности SEQ ID NO: 12, имеющей замену, делецию или добавление от 1 до 4 аминокислот, кроме аминокислот в положениях 1, 2, 3, 4, 6, 11, 12, 14, 19, 26, 30, 33 и 34, и обладающий свойством связывания урацила, где полипептид представлен Формулой 1, приведенной ниже; и
(U-3) полипептид, имеющий идентичность последовательности по меньшей мере 90% с последовательностью SEQ ID NO: 12, при условии, что аминокислоты в положениях 1, 2, 3, 4, 6, 11, 12, 14, 19, 26, 30, 33 и 34 идентичны, и обладающий свойством связывания урацила, где полипептид представлен Формулой 1, приведенной ниже,
(Спираль А)-Х-(Спираль В)-L, Формула 1
в Формуле:
Спираль A является группой длиной 12 аминокислот, способной образовывать структуру α-спирали, и представлена формулой 2
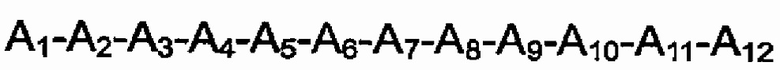 , Формула 2
, Формула 2
где в формуле 2 A1-A12 независимо являются аминокислотой;
X не существует или является группой длиной от 1 до 9 аминокислот;
Спираль B является группой длиной от 11 до 13 аминокислот, способной образовывать структуру α-спирали; и
L является группой длиной от 2 до 7 аминокислот, представленной формулой 3
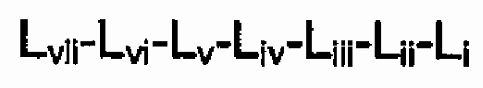 , Формула 3
, Формула 3
где в формуле 3 аминокислоты пронумерованы «i» (-1), «ii» (-2) и так далее со стороны С-конца,
при условии, что Liii-Lvii могут не существовать,
где n равно 7-14.
5. Слитый белок, специфически связывающийся с молекулой РНК-мишенью в клетках, где указанный слитый белок содержит белок по п. 4 и по меньшей мере один белок, выбранный из группы, состоящей из флуоресцентного белка, сигнального пептида ядерной локализации и белка-метки.
6. Способ контроля сплайсинга РНК, где способ включает:
добавление раствора, содержащего белок по п. 4 или слитый белок по п. 5, к образцу, содержащему РНК-мишень, и
культивирование репортера сплайсинга с белком по п. 4 или слитым белком по п. 5,
добавление РНК в культуру для получения реакционной смеси,
подвергание реакционной смеси электрофорезу и
рассчитывание интенсивности полос,
где осуществление способа исключено на индивидууме человека или зародышевой клетке человека.
7. Нуклеиновая кислота, кодирующая белок по п. 1 или 2, где указанная нуклеиновая кислота содержит нуклеотидную последовательность, кодирующую белок по п. 1 или 2.
8. Нуклеиновая кислота, кодирующая белок по п. 4, где указанная нуклеиновая кислота содержит нуклеотидную последовательность, кодирующую белок по п. 4.
9. Экспрессионный вектор, содержащий нуклеиновую кислоту по п. 7 или 8.
10. Клетка для получения белка по п. 1 или 4, где указанная клетка содержит вектор по п. 9 и не является клеткой человека.
11. Способ манипулирования РНК, где способ содержит приведение в контакт РНК-мишени с белком по п. 4 или слитым белком по п. 5, таким образом осуществляя модификацию сплайсинга РНК-мишени,
где осуществление способа исключено на индивидууме человека или зародышевой клетке человека.
12. Способ получения организма, не являющегося человеком, включающий осуществление способа манипулирования по п. 11 в организме, не являющемся человеком.
13. Способ выявления РНК, где способ включает:
добавление раствора, содержащего белок по п. 4 или слитый белок по п. 5, к образцу, содержащему РНК-мишень, и
выявление или количественное измерение указанного белка, который связан с РНК-мишенью,
где осуществление способа исключено на индивидууме человека или зародышевой клетке человека.
| SHEN C | |||
| et al., Structural basis for specific single-stranded RNA recognition by designer pentatricopeptide repeat proteins, NATURE COMMUNICATIONS, 2016, v | |||
| Способ восстановления хромовой кислоты, в частности для получения хромовых квасцов | 1921 |
|
SU7A1 |
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| US 2003126646 А1, 03.07.2003 | |||
| Кипятильник для воды | 1921 |
|
SU5A1 |
| БД "UniProtKB/TrEMBL" последовательность под | |||
Авторы
Даты
2025-01-30—Публикация
2020-05-29—Подача