ОБЛАСТЬ ИЗОБРЕТЕНИЯ
Настоящая заявка относится к области биотехнологии и, в частности, к композиции, набору и применению для выявления колоректального рака.
УРОВЕНЬ ТЕХНИКИ
Колоректальный рак, который преимущественно поражает толстую и прямую кишку в нижнем отделе желудочно-кишечного тракта, представляет собой распространенную злокачественную опухоль. Согласно последним статистическим данным, он занимает третье место в мире среди новых случаев рака у мужчин и второе - у женщин, а также он является четвертой ведущей причиной смерти от рака у мужчин и третьей - у женщин (Bray et al., 2018). В Китае колоректальный рак занимает третье место по количеству новых случаев рака (380000 человек) и пятое по числу умерших (187000 человек) (Zheng Rongshou et al., 2019). Таким образом, существует острая необходимость предупреждения и лечения колоректального рака. Колоректальный рак развивается медленно и обычно проходит через процесс образования полипа, аденомы и, в конечном итоге, злокачественной опухоли. Это может занять от 5 до 10 лет, прежде чем аденома разовьется в рак. Раннее вмешательство на стадиях колоректального рака может значительно снизить смертность. Пятилетняя выживаемость пациентов с колоректальным раком I стадии может превышать 90 %, тогда как пятилетняя выживаемость пациентов с IV стадией - менее 20 % (Marzieh Araghi et al., 2020).
Традиционные методы раннего скрининга колоректального рака включают колоноскопию, анализ кала на скрытую кровь и определение онкомаркеров CEA и CA19-9. Однако все эти методы имеют определенные ограничения. Колоноскопия представляет собой золотой стандарт диагностики колоректального рака. Посредством введения оптического прицела и источника света через задний проход врач может визуализировать прямую, сигмовидную и другие части толстой кишки в режиме реального времени на мониторе. Колоноскопия обеспечивает четкую и прямую визуализацию, позволяя наблюдать различные поражения, такие как рак, полипы, язвы и кровотечения. Кроме того, она позволяет удалять полипы во время процедуры. Однако колоноскопия представляет собой инвазивную процедуру, которая требует сложной подготовки, включая ограничения в диете и подготовку кишечника. Комплаентность среди лиц, проходящих колоноскопию, часто бывает низкой. Кроме того, для ее проведения требуется специальное оборудование и персонал, с обученными специалистами и анестезиологами в условиях стационара. Некоторые субъекты могут испытывать дискомфорт и риск осложнений (3-5 случаев/1000 человек). Анализ кала на скрытую кровь представляет собой неинвазивный способ выявления наличия компонентов крови (гемоглобина) в кале, указывающий на возможное желудочно-кишечное кровотечение и оценивающий риск развития колоректального рака. Такой способ является быстрым и простым, а комплаентность обследуемого - высокой. Однако на это выявление могут повлиять ложноположительные результаты из-за употребления в пищу продуктов печени или крови, а также зеленых листовых овощей. Кроме того, чувствительность является низкой для ранних стадий колоректального рака с минимальным кровотечением, что может привести к пропуску диагноза. Маркеры опухолей, такие как СЕА, представляют собой маркеры широкого спектра действия с низкой специфичностью, которые могут давать больше ложноположительных результатов, а также их чувствительность низкая. Поэтому необходимо разработать точный, простой и экономически эффективный способ раннего скрининга колоректального рака.
Метилирование ДНК представляет собой важный механизм регуляции экспрессии генов, способный модулировать экспрессию генов и подавлять их активность. Оно оказывает существенное влияние на возникновение и развитие опухолей. Аберрантное метилирование генов, связанных с раком, часто происходит на ранних стадиях развития рака, делая метилирование ДНК потенциальным биомаркером для раннего скрининга. Образцы человеческих фекалий включают информацию о ДНК, поскольку эпителий толстой кишки постоянно выделяет клетки в просвет кишечника в ходе клеточного оборота, который несет такую информацию, как метилирование. Обнаружив и проанализировав эту информацию, можно сделать вывод о вероятности заболевания у человека колоректальным раком. Некоторые гены были идентифицированы как маркеры метилирования для выявления колоректального рака в кале, например SFRP2, SEPT9, NDRG4 и SDC2, т. д. (Hannes M Müller et al. 2004; Jie Chen et al. 2019), но их эффективность еще не полностью соответствует клиническим требованиям. Например, чувствительность и специфичность SFRP2 при колоректальном раке составляет всего 77 % (Hannes M Müller et al. 2004).
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
Цель настоящего изобретения - решить по меньшей мере одну из технических проблем в предшествующем уровне техники и предоставить композицию, набор и применение для выявления колоректального рака. Представленную композиция можно применять для выявления колоректального рака с высокой специфичностью и чувствительностью, такой, которая может способствовать клиническому выявлению и обогащению диагностики и выявления колоректального рака.
В частности, в настоящем изобретении предоставлены следующие технические решения.
В первом аспекте настоящее изобретение предоставляет композицию для выявления колоректального рака, которая включает реагенты для выявления статуса метилирования гена ADHFE1, гена PPP2R5C и гена SDC2.
Во втором аспекте настоящее изобретение предоставляет набор для диагностики колоректального рака. Набор включает композицию для выявления колоректального рака в соответствии с первым аспектом, описанным выше.
В третьем аспекте настоящее изобретение предоставляет способ выявления статуса метилирования генов в предоставленном образце. Гены включают ген ADHFE1, ген PPP2R5C и ген SDC2. Способ включает: (1) экстрагирование геномной ДНК из образца; (2) получение продукта конверсии путем осуществления конверсии метилирования на геномной ДНК, где метилированный участок отличается от неметилированного участка после обработки; и (3) применение набора праймеров и зондов для осуществления флуоресцентного количественного ПЦР-выявления продукта конверсии для выявления статуса метилирования гена в образце.
В четвертом аспекте настоящее изобретение предоставляет выделенные последовательности нуклеиновой кислоты, включающие набор праймеров и зонды. В комплект праймера включено: (a) по меньшей мере одну из последовательностей, представленных в SEQ ID NO: 3 - SEQ ID NO: 40; (б) по меньшей мере одну из последовательностей, представленных в SEQ ID NO: 41 - SEQ ID NO: 60; и (в) по меньшей мере одну из последовательностей, представленных в SEQ ID NO: 61 - SEQ ID NO: 98. Зонды включают: по меньшей мере одну из последовательностей, представленных в SEQ ID NO: 100 - SEQ ID NO: 112; по меньшей мере одну из последовательностей, представленных в SEQ ID NO: 113 - SEQ ID NO: 127; и по меньшей мере одну из последовательностей, представленных в SEQ ID NO: 128 - SEQ ID NO: 142.
В пятом аспекте настоящего изобретения предоставляется способ оценивания болезненного состояния колоректального рака у субъекта. Способ включает: а) предоставление образца субъекта, причем образец содержит нуклеиновые кислоты-мишени субъекта, а нуклеиновые кислоты-мишени содержат нуклеиновые кислоты гена ADHFE1, гена PPP2R5C и гена SDC2; б) оценивание статуса метилирования нуклеиновых кислот-мишеней; и в) оценивание болезненного состояния колоректального рака у субъекта на основании статуса метилирования нуклеиновых кислот-мишеней.
Дополнительные аспекты и польза по настоящему изобретению будут частично изложены в последующем описании и частично будут очевидны из описания или могут быть изучены на практике при применении настоящего изобретения.
КРАТКОЕ ОПИСАНИЕ ГРАФИЧЕСКИХ МАТЕРИАЛОВ
Вышеизложенные и/или дополнительные аспекты и польза по настоящему изобретению станут очевидными и легко оценимыми из следующего описания вариантов осуществления, взятого вместе с сопроводительными графическими материалами.
ФИГ. 1 представляет собой схематическое изображение уровней метилирования в ткани колоректальной опухоли и образцах прилегающей нормальной ткани в базе данных Атласа ракового генома (англ.: the Cancer Genome Atlas, TCGA) в соответствии с одним из вариантов осуществления по настоящему изобретению.
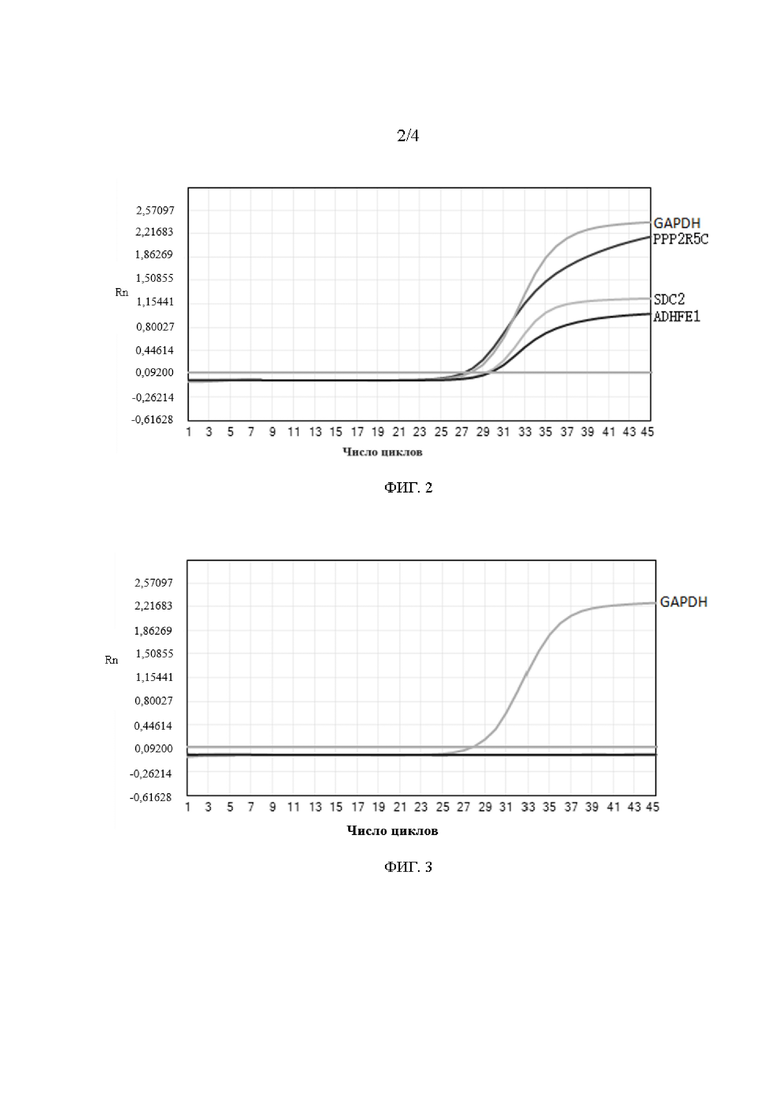
ФИГ. 2 представляет собой график кривой флуоресцентной количественной ПЦР-амплификации положительного стандарта примера 2 по настоящему изобретению.
ФИГ. 3 представляет собой график флуоресцентной кривой количественной ПЦР-амплификации отрицательного стандарта примера 2 по настоящему изобретению.
ФИГ. 4 представляет собой блок-схему выявления набора для диагностики колоректального рака, представленного в примере 3, по настоящему изобретению.
ФИГ. 5 представляет собой ROC-график образцов колоректального рака, обнаруженных для каждого гена в примере 3 по настоящему изобретению.
ПОДРОБНОЕ ОПИСАНИЕ
Теперь будет сделана подробная ссылка на варианты осуществления по настоящему изобретению; следует понимать, что описанные варианты осуществления представляют собой иллюстрацию и предназначены для пояснения, а не ограничения настоящего изобретения. В процессе описания решений, представленных в настоящем изобретении, соответствующие термины в тексте определяли и объясняли. Эти выявления и пояснения предназначены только для удобства понимания решений, и они не должны рассматриваться как ограничения защитных решений настоящего изобретения.
В одном из аспектов настоящего изобретения предоставляется композиция для выявления колоректального рака. Композиция включает реагенты для выявления статуса метилирования гена ADHFE1, гена PPP2R5C и гена SDC2. Композиция, описанная в настоящем документе, не требует, чтобы два или более вещества находились в контакте и перемешивались друг с другом, но подразумевает, что композиция наносится в одной и той же среде в момент применения. Например, при выявлении колоректального рака одновременно выявляют статус метилирования гена ADHFE1, гена PPP2R5C и гена SDC2.
Модификации метилирования гена ADHFE1, гена PPP2R5C и гена SDC2, как упомянуто в настоящем документе, тесно связаны с возникновением и развитием рака и демонстрируют значительные различия между нормальной тканью и опухолевой тканью. Например, ген SDC2, ген ADHFE1 и ген PPP2R5C присутствуют с высоким уровнем метилирования в тканях карциномы. Ген ADHFE1 кодирует трансгидрогеназу, которая вовлечена во внутриклеточный метаболизм и другие физиологические активности, а гиперметилирование ADHFE1, как было показано, способствует пролиферации клеток колоректального рака посредством модуляции прогрессирования клеточного цикла (Hu YH et al. 2019). ADHFE1 гиперметилирован в тканях запущенной аденомы и карциномы (Tae CH et al. 2013), а значительные различия в уровне метилирования ADHFE1 среди нормальных, аденомных и карциномных тканей делают его идеальным маркером при выявлении колоректального рака (Naumov VA et al. 2013. Fan, J et al. 2020). Ген PPP2R5C кодирует регуляторную субъединицу фосфатазы PP2A и представляет собой негативный регулятор роста и пролиферации клеток (Veerle JANSSENS et al. 2001). Сообщалось, что кодируемый им белок может регулировать дефосфорилирование белка p53 в ответ на повреждение ДНК, тем самым ингибируя рост раковых клеток кишечника (Li HH et al. 2007). Статус гиперметилирования гена PPP2R5C тесно относится к возникновению и развитию рака кишечника и может применяться как потенциальный маркер для выявления рака кишечника (Galamb O et al. 2016). Ген SDC2 кодирует внутренний мембранный белок, который вовлечен в клеточное деление, миграцию клеток и интерстициальные взаимодействия (Kim JH et al. 2018). В ряде исследований установили, что уровень метилирования ДНК SDC2, который увеличивается по мере прогрессирования колоректального рака, был значительно выше в тканях колоректального рака, чем в соседних нормальных тканях, и был эффективным маркером для диагностики колоректального рака (Oh TJ et al. 2017).
В настоящее время ген SDC2 стал более надежным стандартом для диагностики и выявления колоректального рака. В настоящем изобретении за счет объединения гена SDC2 с геном ADHFE1 и геном PPP2R5C чувствительность и специфичность выше, чем при выявлении одного гена SDC2. Кроме того, комбинация гена SDC2, гена ADHFE1 и гена PPP2R5C имеет более высокую специфичность и чувствительность для диагностики колоректального рака по сравнению с другими генами, такими как NDRG4, BMP3 и т. д. Например, в настоящем изобретении чувствительность может быть более 85 %, а специфичность может быть более 90 %. Например, чувствительность может составлять более 87 %, даже более 90 %, а специфичность может быть более 90 %, даже более 94 %. Более того, с большей вероятностью будет видна разница между образцами колоректального рака и нормальными образцами.
По меньшей мере в некоторых вариантах осуществления реагенты включают набор праймеров и зондов. Как представленно в настоящем документе, термин «праймер» относится к короткой последовательности нуклеиновой кислоты, имеющей свободную гидроксильную группу на 3'-конце и способной формировать пары оснований с комплементарной матрицы, причем праймер служит источником репликации матричной цепи. В соответствующих буферных и температурных условиях праймер может инициировать синтез ДНК в присутствии различных нуклеозидтрифосфатов и полимеризующих реагентов, таких как ДНК-полимераза или обратная транскриптаза.
Термин «зонд» относится к фрагменту полинуклеотида, такому как РНК или ДНК, который способен специфически связываться с мРНК или комплементарной ДНК (кДНК) конкретного гена и имеет длину от нескольких до нескольких сотен пар оснований. Поскольку зонд помечен, его можно применять для выявления наличия или уровня экспрессии целевой мРНК или кДНК, подлежащей связыванию.
В некоторых вариантах осуществления набор праймеров включает: (a-1) по меньшей мере одну из последовательностей, представленных в SEQ ID NO: 3 - SEQ ID NO: 40 (например, по меньшей мере одну пару этих последовательностей); (б-1) по меньшей мере одну из последовательностей, представленных в SEQ ID NO: 41 - SEQ ID NO: 60 (например, по меньшей мере одну пару этих последовательностей); и (в-1) по меньшей мере одну из последовательностей, представленных в SEQ ID NO: 61 - SEQ ID NO: 98 (например, по меньшей мере одну пару этих последовательностей).
В некоторых вариантах осуществления зонды включают: (a-2) по меньшей мере одну из последовательностей, представленных в SEQ ID NO: 100 - SEQ ID NO: 112; (б-2) по меньшей мере одну из последовательностей, представленных в SEQ ID NO: 113 - SEQ ID NO: 127; и (в -2) по меньшей мере одну из последовательностей, представленных в SEQ ID NO: 128 - SEQ ID NO: 142.
По меньшей мере в некоторых вариантах осуществления набор праймеров включает (a-1) по меньшей мере одну пару последовательностей, представленных в SEQ ID NO: 3 - SEQ ID NO: 40; (б-1) по меньшей мере одну пару последовательностей, представленных в SEQ ID NO: 41 - SEQ ID NO: 60; и (в-1) по меньшей мере одну пару последовательностей, представленных в SEQ ID NO: 61 - SEQ ID NO: 98. По меньшей мере в некоторых вариантах осуществления зонды включают: (a-2) по меньшей мере одну из последовательностей, представленных в SEQ ID NO: 100 - SEQ ID NO: 112; (б-2) по меньшей мере одну из последовательностей, представленных в SEQ ID NO: 113 - SEQ ID NO: 127; и (в -2) по меньшей мере одну из последовательностей, представленных в SEQ ID NO: 128 - SEQ ID NO: 142.
По меньшей мере одна пара вышеупомянутых последовательностей относится к паре праймеров, т.е. к любому прямому праймеру и любому обратному праймеру определенного идентичного гена (т.е. гена ADHFE1, гена PPP2R5C или гена SDC2, упомянутого в настоящем документе). Следует отметить, что когда праймеры и зонды для одного и того же гена применяли в комбинации, любой прямой праймер определенного гена и обратный праймер, расположенный после прямого праймера, а также зонды, расположенные между прямым и обратным праймерами, могли применяться в комбинации для осуществления амплификации и выявления генов, связанных с колоректальным раком. Специалисты в данной области техники могут применять комбинации праймеров и зондов для выравнивания последовательностей с геномом основываясь на последовательностях, представленных в настоящем документе. Например, праймер прямого действия под номером SEQ ID NO: 7 или SEQ ID NO: 11 и обратный праймер под номером SEQ ID NO: 8 или SEQ ID NO: 24 можно применять в комбинации в виде пары праймеров, а также применять их в комбинации с зондами под номером SEQ ID NO: 115, SEQ ID NO: 114, SEQ ID NO: 125 или SEQ ID NO: 126, по желанию.
В некоторых вариантах осуществления набор праймеров дополнительно включает последовательности, представленные в SEQ ID NO: 1 - SEQ ID NO: 2, и зонды дополнительно включают последовательность, представленную в SEQ ID NO: 99. Последовательности, представленные в SEQ ID NO: 1 - SEQ ID NO: 2 или SEQ ID NO: 99 можно применять для ПЦР-амплификации и выявления внутреннего эталонного гена GAPDH.
Соответствующие последовательности перечислены в таблице 1. Наборы праймеров и зонды разработаны с учетом модифицированных метилированием последовательностей генов ADHFE1, PPP2R5C и SDC2, и они могут специфически распознавать модифицированные метилированием последовательности. Ген GAPDH применяли в виде внутреннего эталона, на амплификацию внутреннего эталонного гена не влияла модификация метилирования, позволяющая измерить количество матрицы в образце. Рассчитывая разницу между Ct целевого гена и Ct внутреннего эталонного гена, степень метилирования образца можно определить, и это полезно для выявления колоректального рака. 5'-конец упомянутого зонда можно связать с флуорофором FAM, VIC, ROX и CY5, а 3'-конец можно связать с гасителем, таким как MGB. Флуорофор и гаситель обычно применяют в данной области техники.
В некоторых вариантах осуществления реагенты для выявления гена ADHFE1 включают последовательности, представленные в SEQ ID NO: 7, SEQ ID NO: 8 и SEQ ID NO: 127; реагенты для выявления гена PPP2R5C включают последовательности, представленные в SEQ ID NO: 53, SEQ ID NO: 54 и SEQ ID NO: 139; реагенты для выявления гена SDC2 включают последовательности, представленные в SEQ ID NO: 89, SEQ ID NO: 90 и SEQ ID NO: 101; а композиция дополнительно включает реагент для выявления гена GAPDH, причем реагент для выявления гена GAPDH включает последовательности, представленные в SEQ ID NO: 1, SEQ ID NO: 2 и SEQ ID NO: 99.
Праймеры или зонды, упомянутые выше, можно синтезировать искусственно, например, с помощью синтеза твердофазного носителя на основе фосфонамидита или других способов, обычно применяемых в соответствующей области техники.
По меньшей мере в некоторых вариантах осуществления композиция включает по меньшей мере одно, выбранное из ПЦР-буфера, иона соли, дНТФ, ДНК-полимеразы или нуклеиновой кислоты-мишени. Вышеупомянутые нуклеиновые кислоты-мишени можно экстрагировать из образца с помощью некоторых коммерчески доступных наборов. Нуклеиновая кислота-мишень представляет собой последовательность нуклеиновой кислоты, содержащую гены ADHFE1, PPP2R5C и SDC2.
По меньшей мере в некоторых вариантах осуществления вышеупомянутое флуоресцентное количественное ПЦР-выявление осуществляли при 95°C в течение 10 минут, при 95°C в течение 15 секунд, при 55°C в течение 30 секунд и при 72°C в течение 30 секунд, всего для 45 циклов.
По меньшей мере в некоторых вариантах осуществления соотношение масс набора праймеров и зондов представляет собой 1: 1: 1: 1. При этом можно получить идеальный результат выявления.
В другом аспекте настоящего изобретения предоставляется набор для диагностики колоректального рака. В набор включена композиция как описано выше. Набор, представленный настоящим изобретением, может специфически выявить модификацию метилирования генов ADHFE1, PPP2R5C и SDC2 в образце, тем самым оценивая риск колоректального рака у субъекта. В настоящем изобретении комбинированное определение статуса метилирования трех генов, т.е. ADHFE1, PPP2R5C и SDC2, может эффективно улучшить производительность выявления по сравнению с выявлением метилирования одного гена.
По меньшей мере в некоторых вариантах осуществления набор дополнительно включает по меньшей мере один из следующих реагентов: реагент для экстракции ДНК, реагент для конверсии метилирования ДНК и реагент для очистки нуклеиновой кислоты. Реагент для экстракции ДНК можно применять для получения ДНК в образце. Реагент для конверсии метилирования ДНК может конвертировать цитозин в ДНК в урацил. Реагент для очистки нуклеиновой кислоты можно применять для получения очищенных нуклеиновых кислот, которые затем можно применять в последующих реакционных процессах.
Вышеупомянутый набор можно также применять, как описано ниже, для выявления статуса метилирования гена в образце или оценивания наличия колоректального рака у субъекта. С этой целью в настоящем изобретении дополнительно предоставляется применение реагента при изготовлении набора для выявления статуса метилирования гена в образце. Настоящее изобретение дополнительно предоставляет применение реагента при изготовлении набора для оценивания состояния колоректального рака у субъекта.
В еще одном аспекте настоящее изобретение дополнительно предоставляет способ выявления статуса метилирования генов, включая ген ADHFE1, ген PPP2R5C и ген SDC2, в образце. Способ включает: (1) экстрагирование геномной ДНК из образца; (2) получение продукта конверсии путем превращения геномной ДНК с помощью обработки метилированием; и (3) осуществление с помощью набора праймеров и зондов флуоресцентного количественного ПЦР-выявления продукта конверсии для выявления статуса метилирования гена в образце.
Как правило, статус метилирования относится к наличию или отсутствию 5-метилцитозина в одном или более CpG-динуклеотидах в последовательности ДНК. Способ выявления статуса метилирования генов в образце, представленный в настоящем изобретении, может применяться в качестве точного, простого и экономичного средства для раннего скрининга колоректального рака, тем самым увеличивая уровень выявления колоректального рака, особенно на ранних стадиях рака кишечника, в популяции с высоким риском развития колоректального рака и в популяции, проходящей общий медицинский осмотр. Таким образом, коэффициент выживаемости пациентов с колоректальным раком может быть увеличен, а большинство медицинских расходов и медицинское бремя снижены.
Образец не особо ограничен в настоящем документе, и образец может быть биологическим образцом ex vivo, содержащим нуклеиновые кислоты-мишени, включая, помимо прочего, по меньшей мере один из образцов ткани, крови, слюны, секрета или выделений. Выделения могут быть образцом мочи, пота или слезы. В некоторых предпочтительных вариантах выделения представляют собой образец фекалий. Поскольку образец фекалий содержит эксфолиативные клетки кишечника и имеет неинвазивные характеристики для применения при выявлении, образцы фекалий могут служить предпочтительным тест-объектом.
Вышеупомянутой конверсионной обработкой метилирования могут быть некоторые химические агенты, например бисульфит натрия. Как пример те же коммерчески доступные наборы можно применять для осуществления конверсионной обработки метилирования, например, применяя набор EZ DNA Methylation-Gold Kit для осуществления бисульфитной конверсии ДНК. Полученный продукт можно подвергнуть очистке на колонке и повторному растворению с помощью коммерческого набора для получения очищенного продукта.
В другом аспекте настоящего изобретения дополнительно предоставляются выделяемые последовательности нуклеиновой кислоты, включающие набор праймеров и зондов. В комплект праймера включено: (a) по меньшей мере одну пару последовательностей, представленных в SEQ ID NO: 3 - SEQ ID NO: 40; (б) по меньшей мере одну пару последовательностей, представленных в SEQ ID NO: 41 - SEQ ID NO: 60; и (в) по меньшей мере одну пару последовательностей, представленных в SEQ ID NO: 61 - SEQ ID NO: 98. Зонды включают: по меньшей мере одну из последовательностей, представленных в SEQ ID NO: 100 - SEQ ID NO: 112; по меньшей мере одну из последовательностей, представленных в SEQ ID NO: 113 - SEQ ID NO: 127; и по меньшей мере одну из последовательностей, представленных в SEQ ID NO: 128 - SEQ ID NO: 142.
Настоящее изобретение дополнительно предоставляет способ для оценивания болезненного состояния колоректального рака у субъекта. Способ включает: а) предоставление образца субъекта, причем образец содержит нуклеиновые кислоты-мишени субъекта, а нуклеиновые кислоты-мишени включают нуклеиновые кислоты гена ADHFE1, гена PPP2R5C и гена SDC2; б) оценивание статуса метилирования нуклеиновых кислот-мишеней; и в) оценивание болезненного состояния колоректального рака у субъекта основываясь на статусе метилирования нуклеиновых кислот-мишеней. Реагентами для оценивания статуса метилирования нуклеиновых кислот-мишеней могут быть химические реагенты, такие как сульфит или гидросульфит, бисульфит, например, гидросульфит натрия. В нуклеиновых кислотах-мишенях, обработанных этими химическими реагентами, неметилированный цитозин может конвертироваться в урацил, тогда как метилированный цитозин не подвержен такой конверсии. Затем различия конвертированных последовательностей анализировали различными методами, такими как секвенирование, ПЦР, анализ кривой плавления с высоким разрешением и т. д., в частности, с помощью флуоресцентной количественной ПЦР для быстрой и легкой идентификации. Реагентами для оценивания статуса метилирования нуклеиновых кислот-мишеней также могут быть некоторые биологические реагенты, такие как полипептиды или ферменты, такие как некоторые чувствительные к метилированию рестрикционные ферменты.
Субъект, упомянутый в настоящем документе, представляет собой человека, например, пациента, нуждающегося в диагностике. Биологические образцы, полученные от субъекта, включают, помимо прочего, по меньшей мере один из секретов или выделений, таких как образцы тканей, образцы крови, образцы слюны и т.п. Выделения могут быть образцом мочи, пота или слезы. Кроме того, поскольку образец фекалий содержит эксфолиативные клетки кишечника и имеет неинвазивные характеристики для применения при выявлении, образцы фекалий могут служить предпочтительным тест-объектом.
По меньшей мере в некоторых вариантах осуществления субъект представляет собой млекопитающее. По меньшей мере в некоторых вариантах осуществления статус метилирования нуклеиновой кислоты-мишени оценивали на шаге б) с помощью вышеупомянутого набора праймеров и зонда.
Способ дополнительно включает: выделение нуклеиновых кислот-мишеней из образца. Нуклеиновые кислоты-мишени можно выделить из образца с помощью коммерчески доступных наборов, известных в данной области техники.
Способ дополнительно включает: амплификацию нуклеиновых кислот-мишеней. Обычные реагенты для амплификации нуклеиновых кислот могут быть применены. Реагенты для амплификации нуклеиновых кислот могут включать фермент, например фермент, который можно применять в реакции полинуклеотидной амплификации, такой как флуоресцентная количественная полимеразная реакция (кПЦР).
Способ дополнительно включает: оценивание статуса метилирования нуклеиновых кислот-мишеней для получения показателя метилирования; и сравнение показателя метилирования с эталонным значением для выявления статуса метилирования образца. В некоторых вариантах осуществления способ включает: оценивание статуса метилирования нуклеиновой кислоты-мишени с помощью химического реагента. В соответствии с предпочтительным вариантом осуществления по настоящему изобретению, химический реагент включает гидросульфит или сульфит. В некоторых предпочтительных вариантах осуществления эталонное значение получали из внутреннего эталонного гена GAPDH. Амплифицированные значения Ct генов ADHFE1, PPP2R5C и SDC2 сравнивали с амплифицированным значением Ct внутреннего эталонного гена GAPDH для выявления статуса метилирования гена образца основываясь на пороговом значении. Вышеупомянутое пороговое значение можно установить основываясь на анализе большого количества клинических образцов известной патологической информации. Вышеупомянутый показатель метилирования также может быть любым другим общим показателем метилирования, таким как частота метилирования, нагрузка гаплотипов метилирования и т. д.
По меньшей мере в некоторых вариантах осуществления способ дополнительно включает: определение того, удовлетворяет ли содержание ДНК обнаружению, с помощью внутреннего эталонного гена, и определение статуса метилирования с помощью разницы (ΔCt) между значением Ct целевого гена и значением Ct внутреннего эталонного гена.
По меньшей мере в некоторых вариантах осуществления способ дополнительно включает: оценивание болезненного состояния колоректального рака у субъекта при помощи метода флуоресцентной количественной ПЦР с помощью GAPDH в виде внутреннего эталонного гена; определение, когда значение Ct GAPDH больше 37, неудовлетворительного контроля качества; определение, когда значения Ct генов ADHFE1, PPP2R5C и SDC2, болезненного состояния колоректального рака у субъекта в виде отрицательного; и вычисляем, когда значения Ct генов ADHFE1, PPP2R5C и SDC2 составляют ≤ 37, разницу ΔCt между амплифицированными значениями Ct ADHFE1, PPP2R5C и SDC2 генов и амплифицированное значение Ct внутреннего эталона гена GAPDH, и оценивание состояния заболевания колоректальным раком у субъекта основываясь на результатах ROC-кривой и результатах AUC ΔCt генов ADHFE1, PPP2R5C и SDC2.
Способ дополнительно включает: определение того, когда ΔCt(ADHFE1) ≤ 8, ΔCt(PPP2R5C) ≤ 5 и ΔCt(SDC2) ≤ 12, болезненное состояние колоректального рака у субъекта как положительное.
Способ дополнительно включает: оценивание вероятности колоректального рака основываясь на статусе метилирования субъекта, тем самым помогая в диагностике и скрининге опухолей, чтобы обеспечить субъекту лечение как можно раньше. По меньшей мере в некоторых вариантах осуществления лечение включает по меньшей мере одно из химиотерапии, лучевой терапии, иммунотерапии, клеточной терапии и хирургического вмешательства.
Варианты осуществления по настоящему изобретению были объяснены со ссылкой на следующие примеры. Специалистам в данной области техники будет понятно, что приведенные ниже примеры представляют собой лишь иллюстрацию и их не следует рассматривать как ограничение объема настоящего изобретения. Конкретные методы или условия, которые не указаны в примерах, должны соответствовать тем, которые описаны в литературе в смежных областях техники или в соответствии с описанием продукта. Применяемые реактивы и инструменты без указания производителя представляют собой обычные продукты, которые можно получить коммерческим путем.
Пример 1
Заявитель идентифицировал нуклеиновые кислоты-мишени для диагностики колоректального рака в соответствии со следующими способами и процедурами.
Во-первых, Заявитель сравнивал значимые различия в уровнях метилирования различных генов в опухолевых и неопухолевых образцах в базе данных Атласа ракового генома (англ.: the Cancer Genome Atlas, TCGA) и отбирал гены-кандидаты для выявления метилирования, а именно CDH4, ZNF132, PPP2R5C, LONRF2, ADHFE1, SDC2, NDRG4 и BMP3.
Во-вторых, специфические праймеры и зонды разрабатывали для этих генов-кандидатов. В процессе разработки праймеров и зондов Заявитель выяснил, что в результате метилирования нуклеиновых кислот-мишеней большинство цитозинов были конвертированы в урациловые основания после бисульфитного конвертирования выявляемой последовательности генома, а сама последовательность генома имеет низкую сложность, таким образом, единичные основания могут постоянно присутствовать при разработке праймеров и зондов. Кроме того, температура отжига обычно была низкой. В праймерах было несколько областей, которые можно было разработать.
Затем, основываясь на праймерах и зондах, полученных в результате вышеописанного проектирования, проводили предварительный скрининг генов-кандидатов с помощью небольшого количества опухолевых/неопухолевых образцов. Результаты чувствительности/специфичности каждого гена были следующими: CDH4 (61,5%/61,5%), ZNF132 (50%/100%), PPP2R5C (78,9%/100%), LONRF2 (77,8%/72%), ADHFE1 (100%/91,3%), SDC2 (88,9%/100%), NDRG4 (70%/95%) и BMP3 (65%/90%). Среди них чувствительность/специфичность BMP3, NDRG4, ADHFE1, PPP2R5C и SDC2 была лучше.
Кроме того, сравнивая данные метилирования вышеуказанных пяти генов-кандидатов с лучшей чувствительностью/специфичностью в базе данных TCGA выявили, что все гены ADHFE1, PPP2R5C и SDC2 демонстрируют более низкий фон в тканях паракарциномы, чем гены BMP3 и NDRG4, а также имеют более значительную разницу в тканях рака и паракарциномы, что показано на ФИГ. 1.
Поэтому Заявитель окончательно определил гены ADHFE1, PPP2R5C и SDC2 как нуклеиновые кислот-мишени для диагностики колоректального рака и Заявитель дополнительно разработал и оптимизировал специфические последовательности праймеров и зондов для вышеуказанных генов-мишеней, как указано в таблице 1.
Пример 2
Заявитель применял праймеры и зонды к нуклеиновым кислотам-мишеням (генам ADHFE1, PPP2R5C, SDC2), полученным в примере 1 для диагностики колоректального рака, для амплификации нуклеиновых кислот-мишеней с помощью флуоресцентной количественной ПЦР в положительных или негативных клеточных линиях для проверки эффектов праймеров и зондов.
Шаг 1. Сбор образцов: положительная стандартная ДНК (стандартный набор метилированной ДНК человека CpGenome, Sigma-Aldrich, S8001M); отрицательная стандартная ДНК (стандартный набор неметилированной ДНК человека CpGenome, Sigma-Aldrich, S8001U)
Шаг 2. Модификация ДНК: отбирали по 10 нг каждой из указанных стандартных ДНК, применяли набор EZ DNA Methylation-Gold Kit (ZYMO RESEARCH) для осуществления бисульфитной конверсии ДНК; после очистки колонки 33 мкл воды NF применяли для повторного растворения с последующим применением.
Шаг 3. ПЦР-выявление
Например, применяя праймеры и зонды, перечисленные в таблице 1, флуоресцентно-количественное ПЦР-выявление осуществляли в соответствии со следующей системой ПЦР и процедурой реакции ПЦР. Праймеры и зонды для ADHFE1 выбирали из SEQ ID NO: 7, SEQ ID NO: 8 и SEQ ID NO: 127. Праймеры и зонды для PPP2R5C выбирали из SEQ ID NO: 53, SEQ ID NO: 54 и SEQ ID NO: 139. Праймеры и зонды для SDC2 выбирали из SEQ ID NO: 89, SEQ ID NO: 90 и SEQ ID NO: 101. Праймеры и зонды для внутреннего эталонного гена GAPDH выбирали из SEQ ID NO: 1, SEQ ID NO: 2 и SEQ ID NO: 99.
5'-концом зонда SEQ ID NO: 127 был FAM, а 3'-концом - BHQ1;
5'-концом зонда SEQ ID NO: 139 был CY5, а 3'-концом - BHQ3;
5'-концом зонда SEQ ID NO: 101 был VIC, а 3'-концом - BHQ1; и
5'-концом зонда SEQ ID NO: 99 был ROX, а 3'-концом - MGB.
(1) Система ОППО (англ.: total pathologic complete response, TPCR) показана в Таблице 2:
(2) Процедура проведения ПЦР-реакции приведена в табл. 3:
[Таблица 3] Процедура проведения ПЦР-реакции
Шаг 4. Анализ результатов: рассчитывали значение разницы (Δзначение ΔCt) между амплифицированными значениями Ct генов ADHFE1, PPP2R5C и SDC2 и амплифицированным значением Ct внутреннего эталонного гена GAPDH, а также оценивали, является ли степень метилирования гена образца нормальной, основываясь на пороге суждения, который устанавливали основываясь на анализе большого количества клинических образцов с известной патологоанатомической информацией.
Результаты амплификации вышеуказанных праймеров и зондов показаны на ФИГ. 2 и ФИГ. 3.
Пример 3
Настоящий Заявитель спроектировал и разработал набор для диагностики колоректального рака. Набор включает специфические праймеры и зонды для выявления статуса метилирования гена ADHFE1, гена PPP2R5C и гена SDC2. Специфические праймеры, содержащиеся в различных наборах, выбирали из праймеров, перечисленных в таблице 1, а зонды - из зондов, перечисленных в таблице 1. Каждый набор включает по меньшей мере одну пару прямых и обратных праймеров для специфического выявления гена ADHFE1, по меньшей мере одну пару прямых и обратных праймеров для специфического выявления гена PPP2R5C и по меньшей мере одну пару прямых и обратных праймеров для специфического выявления гена SDC2. Кроме того, набор может дополнительно включать по меньшей мере один зонд для выявления гена ADHFE1, по меньшей мере один зонд для выявления гена PPP2R5C и по меньшей мере один зонд для выявления гена SDC2, если это необходимо.
Разработанный набор может дополнительно включать прямые и обратные праймеры и зонды для выявления внутреннего эталонного гена, например GAPDH, если это необходимо.
5'-конец зонда можно присоединить к флуорофору, такому как FAM, VIC, ROX или CY5, а 3'-конец зонда - к гасителю, такому как MGB.
Последовательности праймеров и зондов в наборе можно обеспечивать в форме порошка.
Кроме того, набор может дополнительно содержать реакционный ПЦР-буфер, полимеразу, реагент для конверсии метилирования ДНК и т.д., если необходимо, тем самым облегчая диагностику колоректального рака специалистами в данной области техники с помощью способа по настоящему изобретению.
Пример 4
Заявитель применял набор, представленный в примере 3, для диагностики колоректального рака, в котором метилирование образца обнаружили с помощью следующего способа, включающего шаги, проиллюстрированные на ФИГ. 4.
1. Сбор образцов
Всего собрали 201 образец колоректального рака, 184 здоровых контрольных образца, 4 образца аденомы и 7 образцов полипов.
2. Экстракция ДНК образца
Собранные образцы подвергали экстракции ДНК с помощью коммерческого набора для экстракции ДНК из образцов фекалий.
3. Модификация ДНК
ДНК подвергали бисульфитной конверсии с помощью набора EZ DNA Methylation-Gold Kit (ZYMO RESEARCH), очищали на колонке и повторно растворяли для дальнейшего применения.
4. ПЦР-выявление
В этом примере, праймеры и зонды для ADHFE1 выбирали из SEQ ID NO: 7, SEQ ID NO: 8 и SEQ ID NO: 127; праймеры и зонды для PPP2R5C выбирали из SEQ ID NO: 53, SEQ ID NO: 54 и SEQ ID NO: 139; праймеры и зонды для SDC2 выбирали из SEQ ID NO: 89, SEQ ID NO: 90 и SEQ ID NO: 101; праймеры и зонды для внутреннего эталонного гена GAPDH выбирали из SEQ ID NO: 1, SEQ ID NO: 2 и SEQ ID NO: 99. Собранные образцы подвергали флуоресцентному количественному ПЦР-определению в соответствии с системой ПЦР и условиями реакции, как описано в Примере 2.
5. Результаты анализов
Выявление осуществляли с помощью прибора SLAN 96S кПЦР. Исходные и пороговые значения устанавливали по умолчанию. Пороговое значение величины Ct каждого гена составляло 37.
Если значение Ct для GAPDH превышало 37, то это расценивали как нарушение контроля качества.
Если генов ADHFE1, PPP2R5C и SDC2 было больше 37, это расценивали как отрицательный результат.
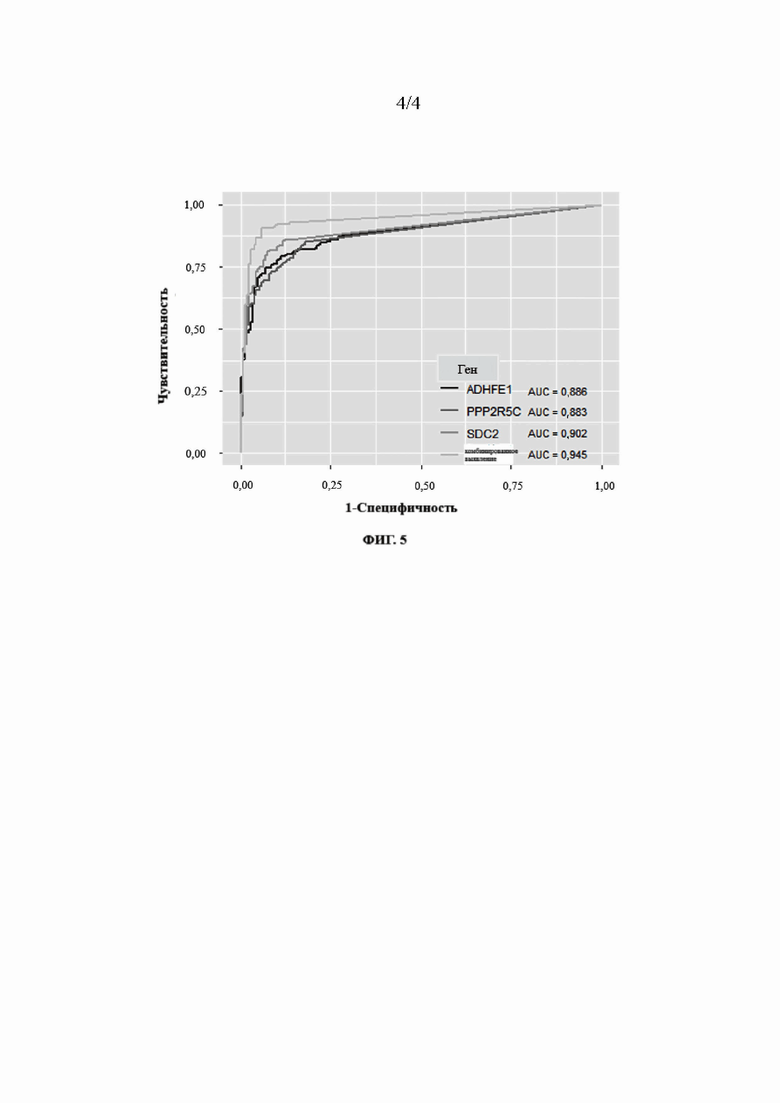
Если значение Ct для генов ADHFE1, PPP2R5C и SDC2 было ≤ 37, разница (значение ΔCt) между амплифицированными значениями Ct для генов ADHFE1, PPP2R5C и SDC2 и амплифицированного значения Ct для генов GAPDH рассчитывали. Программное обеспечение SPSS применяли для построения ROC-кривых значений ΔCt трех обнаруживаемых генов. Кривые ROC показаны на ФИГ. 5, а производительность соответствующих генов и площадь под кривой (AUC) приведены в таблице 4.
[Таблица 4] Производительность каждого гена и площадь под кривой (AUC)
Чтобы еще больше улучшить производительность набора, значения ΔCt трех генов интегрировали для анализа, и AUC могла достигать 0,945. В конечном итоге, ΔCt(ADHFE1) ≤ 8, ΔCt(PPP2R5C) ≤ 5, and ΔCt(SDC2) ≤ 12 были определены положительными для генов. Если какой-либо ген был положительным, образец считали положительным. При этом условии, когда специфичность составляла 94,02%, чувствительность могла достигать 87,56%.
Среди них чувствительность применяли для обозначения процента положительных образцов, диагностированных как колоректальный рак при клинико-патологическом диагнозе, или пациентов, которым диагностирован колоректальный рак в результате выявления.
Специфичность относится к количеству образцов, диагностированных как нормальные при клинико-патологическом диагнозе, или к количеству пациенту, в конечном итоге диагностированных как нормальные.
Приведенные выше результаты показывают, что благодаря комбинированному выявлению множества генов настоящее изобретение может обеспечить более высокую эффективность выявления, чем выявление одного гена.
В описании ссылки на описания терминов «один вариант осуществления», «некоторые варианты осуществления», «примеры», «конкретные примеры» или «некоторые примеры» и т. д. означают, что конкретный признак, структура, материал или характеристика, описанные в связи с вариантом осуществления или примером, включен по меньшей мере в один вариант осуществления или пример настоящего изобретения. В этом описании схематические изображения вышеупомянутых терминов не обязательно относятся к одному и тому же варианту осуществления или примеру. Кроме того, описанные конкретные признаки, структуры, материалы или характеристики можно объединить любым подходящим способом в любом одном или нескольких вариантах осуществления или примерах. Кроме того, специалисты в данной области техники могут осуществить комбинации и комбинации различных вариантов осуществления или примеров и особенностей различных вариантов осуществления или примеров, описанных в настоящем описании, не выходя за объем настоящего изобретения.
Варианты осуществления по настоящему изобретению проиллюстрированы и описаны выше. Следует понимать, что описанные выше варианты осуществления изобретения являются иллюстративными, а не ограничивающими, и специалисты в данной области техники могут вносить в них изменения, модификации, замены и дополнения, не выходя за объем настоящего изобретения.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> BGI GENOMICS CO., LTD.
<120> КОМПОЗИЦИЯ, НАБОР И ПРИМЕНЕНИЕ ДЛЯ ВЫЯВЛЕНИЯ КОЛОРЕКТАЛЬНОГО РАКА
<130> BI3210459P
<160> 142
<170> PatentIn версия 3.5
<210> 1
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
< 223> Прямой праймер GAPDH 1
<400> 1
tttaggagtg agtggaagat aga 23
<210> 2
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
< 223> обратный праймер GAPDH 1
<400> 2
aaaccacacc atcctaatta cct 23
<210> 3
<211> 17
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер ADHFE1 1
<400> 3
tcgttggggt agttggc 17
<210> 4
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер ADHFE1 1
<400> 4
cctatctaaa cctcaaacca atcg 24
<210> 5
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер ADHFE1 2
<400> 5
gttttgggtt tttatcgcgc 20
<210> 6
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер ADHFE1 2
<400> 6
gccctaaaaa actacatcgc g 21
<210> 7
<211> 15
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер ADHFE1 3
<400> 7
gatggtgcga gcgtc 15
<210> 8
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер ADHFE1 3
<400> 8
ctatctaaac ctcaaaccaa tcg 23
<210> 9
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер ADHFE1 4
<400> 9
ggtaatttta agggtggatg 20
<210> 10
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер ADHFE1 4
<400> 10
ctcaaaacca ttttcccac 19
<210> 11
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер ADHFE1 5
<400> 11
aagtaaggag atttaaggta g 21
<210> 12
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер ADHFE1 5
<400> 12
ctcaaaacca ttttcccac 19
<210> 13
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер ADHFE1 6
<400> 13
gggttatttg tttaagtttt aagt 24
<210> 14
<211> 16
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер ADHFE1 6
<400> 14
ccccaaatcc tcaaca 16
<210> 15
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер ADHFE1 7
<400> 15
gttgtagttg ttttagtaag t 21
<210> 16
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер ADHFE1 7
<400> 16
ttaaaactta aacaaataac cc 22
<210> 17
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер ADHFE1 8
<400> 17
gattggtttg aggtttaga 19
<210> 18
<211> 16
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер ADHFE1 8
<400> 18
aatcctcttc cctcct 16
<210> 19
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер ADHFE1 9
<400> 19
gattggtttg aggtttaga 19
<210> 20
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер ADHFE1 9
<400> 20
ttacaattac ctcaacaaat ac 22
<210> 21
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер ADHFE1 10
<400> 21
ggtaatttta agggtggatg gtg 23
<210> 22
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер ADHFE1 10
<400> 22
tcacctatct aaacctcaaa cca 23
<210> 23
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер ADHFE1 11
<400> 23
aggtttagat aggtgatttc gc 22
<210> 24
<211> 16
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер ADHFE1 11
<400> 24
aaactacccg cctccc 16
<210> 25
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер ADHFE1 12
<400> 25
ttggcgtttt ggtttttatt tc 22
<210> 26
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер ADHFE1 12
<400> 26
cctatctaaa cctcaaacca atcg 24
<210> 27
<211> 26
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер ADHFE1 13
<400> 27
gatttaaggt agaattcgag gtttac 26
<210> 28
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер ADHFE1 13
<400> 28
cacgaaataa aaaccaaaac g 21
<210> 29
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер ADHFE1 14
<400> 29
cgtagttttt tgggtgtgat tc 22
<210> 30
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер ADHFE1 14
<400> 30
aaaacgatat ccaaattctc cg 22
<210> 31
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер ADHFE1 15
<400> 31
cgtattttgt gatttcgttg tttc 24
<210> 32
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер ADHFE1 15
<400> 32
aaaaattcga aaaatttaaa taccg 25
<210> 33
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер ADHFE1 16
<400> 33
cgtttgttta ttcgtttcgc 20
<210> 34
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер ADHFE1 16
<400> 34
cgccgctaaa acaactaacg 20
<210> 35
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер ADHFE1 17
<400> 35
cgttttgaga ggttgtatcg c 21
<210> 36
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер ADHFE1 17
<400> 36
aaaaattcga aaaatttaaa taccg 25
<210> 37
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер ADHFE1 18
<400> 37
gtttagggcg gtatttaaat ttttc 25
<210> 38
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер ADHFE1 18
<400> 38
atataacccg ctcaaatcct acg 23
<210> 39
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер ADHFE1 19
<400> 39
tgtatcgcgt attttgtgat ttc 23
<210> 40
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер ADHFE1 19
<400> 40
ccgcttactt tcaaaaattc g 21
<210> 41
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер PPP2R5C 1
<400> 41
ggttttcgtt gtttcgtttc 20
<210> 42
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер PPP2R5C 1
<400> 42
ccaaatcgac gtcctccg 18
<210> 43
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер PPP2R5C 2
<400> 43
ggttttaggt cggggtttc 19
<210> 44
<211> 16
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер PPP2R5C 2
<400> 44
cctcctccga actccg 16
<210> 45
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер PPP2R5C 3
<400> 45
tttcggtatg ggttttaggt c 21
<210> 46
<211> 16
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер PPP2R5C 3
<400> 46
cctcctccga actccg 16
<210> 47
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер PPP2R5C 4
<400> 47
cgttgtttcg tttcggagtt c 21
<210> 48
<211> 17
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер PPP2R5C 4
<400> 48
caaatcgacg tcctccg 17
<210> 49
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер PPP2R5C 5
<400> 49
tgtttcggtt ttcgttgttt c 21
<210> 50
<211> 17
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер PPP2R5C 5
<400> 50
caaatcgacg tcctccg 17
<210> 51
<211> 16
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер PPP2R5C 6
<400> 51
ggaaacggga gagcgc 16
<210> 52
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер PPP2R5C 6
<400> 52
cgccgatcta acctcctacg 20
<210> 53
<211> 17
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер PPP2R5C 7
<400> 53
cgggacggaa aggaaac 17
<210> 54
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер PPP2R5C 7
<400> 54
gccgatctaa cctcctacg 19
<210> 55
<211> 17
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер PPP2R5C 8
<400> 55
ggttggagga tgaagtt 17
<210> 56
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер PPP2R5C 8
<400> 56
tccaaccctc tacttaac 18
<210> 57
<211> 16
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер PPP2R5C 9
<400> 57
ggtatgggtt ttaggt 16
<210> 58
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер PPP2R5C 9
<400> 58
actaaaaaat aaaaccccc 19
<210> 59
<211> 16
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер PPP2R5C 10
<400> 59
aagtagaggg ttggag 16
<210> 60
<211> 16
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер PPP2R5C 10
<400> 60
aaaaccctta actccc 16
<210> 61
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер SDC2 1
<400> 61
agtgtagaaa ttaataagtg agagggc 27
<210> 62
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер SDC2 1
<400> 62
ttcctcctcc tacgcctact 20
<210> 63
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер SDC2 2
<400> 63
ataagtgaga gggcgtcgc 19
<210> 64
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер SDC2 2
<400> 64
ttcctcctcc tacgcctact 20
<210> 65
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер SDC2 3
<400> 65
cgttgcggta ttttgtttc 19
<210> 66
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер SDC2 3
<400> 66
aacgaaacct cctacccaa 19
<210> 67
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер SDC2 4
<400> 67
aaattagaaa ttgaatttcg gtacg 25
<210> 68
<211> 31
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер SDC2 4
<400> 68
aactactctc taaaactctt cttactctac t 31
<210> 69
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер SDC2 5
<400> 69
gcgttgggta ggaggtttc 19
<210> 70
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер SDC2 5
<400> 70
atattcccca aaaaccgact act 23
<210> 71
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер SDC2 6
<400> 71
tcgtttaggg tgtttgaagt tac 23
<210> 72
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер SDC2 6
<400> 72
ccaacgaatc tccgatatat actt 24
<210> 73
<211> 17
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер SDC2 7
<400> 73
cgtggtttgg gaaggac 17
<210> 74
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер SDC2 7
<400> 74
cgaaataaaa ccgttccctc 20
<210> 75
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер SDC2 8
<400> 75
ggagtttggg tcgggttc 18
<210> 76
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер SDC2 8
<400> 76
aaatttccaa atcatcccta atctc 25
<210> 77
<211> 28
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер SDC2 9
<400> 77
gggagtgtag aaattaataa gtgagagg 28
<210> 78
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер SDC2 9
<400> 78
ctcctacgcc tactcccg 18
<210> 79
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер SDC2 10
<400> 79
gagttcgagt tttcgagttt gagt 24
<210> 80
<211> 17
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер SDC2 10
<400> 80
ctacccaacg ctcgacg 17
<210> 81
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер SDC2 11
<400> 81
gagttcgagt tttcgagttt gagt 24
<210> 82
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер SDC2 11
<400> 82
cgaaacctcc tacccaacg 19
<210> 83
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер SDC2 12
<400> 83
gagtcggtga gtgggttagg 20
<210> 84
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер SDC2 12
<400> 84
gctcccctat tccctacg 18
<210> 85
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер SDC2 13
<400> 85
atattttaaa cggcgcgc 18
<210> 86
<211> 31
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер SDC2 13
<400> 86
taaatcctac tcaccttaaa cttaataacc t 31
<210> 87
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер SDC2 14
<400> 87
ttgaggaagg gaagaggg 18
<210> 88
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер SDC2 14
<400> 88
caatccccaa atatacaccg 20
<210> 89
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер SDC2 15
<400> 89
tttagggatc ggttgttttt agtc 24
<210> 90
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер SDC2 15
<400> 90
gaattcgtat acgcgaacta cg 22
<210> 91
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер SDC2 16
<400> 91
agtttaaggt gagtaggatt tacgc 25
<210> 92
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер SDC2 16
<400> 92
ttttacccta attacaaaca acgac 25
<210> 93
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер SDC2 17
<400> 93
ggtttttgcg gttagattaa atc 23
<210> 94
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер SDC2 17
<400> 94
aaaaaacgct actcgctaac gt 22
<210> 95
<211> 16
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер SDC2 18
<400> 95
gtagggaata ggggag 16
<210> 96
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер SDC2 18
<400> 96
aaataacttc aaaaacctc 19
<210> 97
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер SDC2 19
<400> 97
gaagaagttg ggagtgat 18
<210> 98
<211> 16
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер SDC2 19
<400> 98
cctcttccct tcctca 16
<210> 99
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд GAPDH 1
<400> 99
cccaaaacac atttcttcca ttc 23
<210> 100
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд SDC2 1
<400> 100
aaacgaagtt ttttgtttag cgtt 24
<210> 101
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд SDC2 2
<400> 101
gaaaaaactt cgttttaccc taattac 27
<210> 102
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд SDC2 3
<400> 102
acaccgaaaa ttacgattta atctaac 27
<210> 103
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд SDC2 4
<400> 103
acaccaaaaa ttacaattta atctaac 27
<210> 104
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд SDC2 5
<400> 104
ctactcgcta acgttaccc 19
<210> 105
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд SDC2 6
<400> 105
ctactcacta acattaccc 19
<210> 106
<211> 16
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд SDC2 7
<400> 106
cctcttccct tcctca 16
<210> 107
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд SDC2 8
<400> 107
taacgccaac gaacaacgct tt 22
<210> 108
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд SDC2 9
<400> 108
ctaaaccaaa ccgcaattct cgata 25
<210> 109
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд SDC2 10
<400> 109
taacaccaac aaacaacact tt 22
<210> 110
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд SDC2 11
<400> 110
ctaaaccaaa ccacaattct caata 25
<210> 111
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд SDC2 12
<400> 111
accccgcgat ccgcgaaaat aaaa 24
<210> 112
<211> 28
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд SDC2 13
<400> 112
cccgcgatcc gcgaaaataa aaacaccg 28
<210> 113
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд ADHFE1 1
<400> 113
tcacctatct aaacctcaaa cca 23
<210> 114
<211> 17
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд ADHFE1 2
<400> 114
cccaacgacg ctcgcac 17
<210> 115
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд ADHFE1 3
<400> 115
caacgacgct cgcaccat 18
<210> 116
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд ADHFE1 4
<400> 116
taaataatac gaacgccgct aaaac 25
<210> 117
<211> 26
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд ADHFE1 5
<400> 117
atacgaacgc cgctaaaaca actaac 26
<210> 118
<211> 17
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд ADHFE1 6
<400> 118
cccaacaaca ctcacac 17
<210> 119
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд ADHFE1 7
<400> 119
caacaacact cacaccat 18
<210> 120
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд ADHFE1 8
<400> 120
taaataatac aaacaccact aaaac 25
<210> 121
<211> 26
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд ADHFE1 9
<400> 121
atacaaacac cactaaaaca actaac 26
<210> 122
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд ADHFE1 10
<400> 122
actcaaaacc attttcccac gaaat 25
<210> 123
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд ADHFE1 11
<400> 123
aacgaaaaac gccactaccc gatacga 27
<210> 124
<211> 28
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд ADHFE1 12
<400> 124
aacgccacta cccgatacga actacacc 28
<210> 125
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд ADHFE1 13
<400> 125
aaaaaccaaa acgccaacta cccca 25
<210> 126
<211> 28
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд ADHFE1 14
<400> 126
cccacgaaat aaaaaccaaa acgccaac 28
<210> 127
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд ADHFE1 15
<400> 127
caaaacgcca actaccccaa cgac 24
<210> 128
<211> 16
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд PPP2R5C 1
<400> 128
cgaaccgcgc tctccc 16
<210> 129
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд PPP2R5C 2
<400> 129
tttcctttcc gtcccgtctc g 21
<210> 130
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд PPP2R5C 3
<400> 130
aattaaaacg cgcaacgcaa 20
<210> 131
<211> 16
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд PPP2R5C 4
<400> 131
cgattcgcga acgacc 16
<210> 132
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд PPP2R5C 5
<400> 132
ctttaccccg acctccgcta 20
<210> 133
<211> 16
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд PPP2R5C 6
<400> 133
caaaccacac tctccc 16
<210> 134
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд PPP2R5C 7
<400> 134
tttcctttcc atcccatctc a 21
<210> 135
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд PPP2R5C 8
<400> 135
aattaaaaca cacaacacaa 20
<210> 136
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд PPP2R5C 9
<400> 136
acacaattca caaacaacca 20
<210> 137
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд PPP2R5C 10
<400> 137
ctttacccca acctccacta 20
<210> 138
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд PPP2R5C 11
<400> 138
ccgcccgcga accgcgctc 19
<210> 139
<211> 28
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд PPP2R5C 12
<400> 139
tacgcgctcc aaccctctac ttaaccgc 28
<210> 140
<211> 26
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд PPP2R5C 13
<400> 140
caaccctcta cttaaccgcc cgcgaa 26
<210> 141
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд PPP2R5C 14
<400> 141
cgttacgcgc tccaaccctc t 21
<210> 142
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> зонд PPP2R5C 15
<400> 142
tacgttacgc gctccaaccc tct 23
<---

Изобретение относится к области молекулярной биологии. Описана композиция для выявления колоректального рака, содержащая реагенты для определения статуса метилирования гена ADHFE1, гена PPP2R5C и гена SDC2, в которой реагенты содержат разработанные олигонуклеотиды. Также описаны способ выявления статуса метилирования генов в образце и способ оценивания болезненного состояния колоректального рака у субъекта для диагностики колоректального рака с применением указанной композиции. Технический результат заключается в разработке композиции, которую можно применять для выявления колоректального рака с высокой специфичностью и чувствительностью, такой, которая может способствовать клиническому выявлению и расширению арсенала средств диагностики и выявления колоректального рака. 4 н. и 9 з.п. ф-лы, 5 ил., 4 табл., 4 пр.
1. Композиция для выявления колоректального рака, содержащая реагенты для определения статуса метилирования гена ADHFE1, гена PPP2R5C и гена SDC2,
в которой:
реагент для выявления гена ADHFE1 содержит последовательности, представленные в SEQ ID NO: 7, SEQ ID NO: 8 и SEQ ID NO: 127;
реагент для выявления гена PPP2R5C содержит последовательности, представленные в SEQ ID NO: 53, SEQ ID NO: 54 и SEQ ID NO: 139; и
реагент для выявления гена SDC2 содержит последовательности, представленные в SEQ ID NO: 89, SEQ ID NO: 90 и SEQ ID NO: 101.
2. Композиция по п. 1, причем:
композиция дополнительно содержит реагент для выявления гена GAPDH, при этом реагент для выявления гена GAPDH содержит последовательности, представленные в SEQ ID NO: 1, SEQ ID NO: 2 и SEQ ID NO: 99.
3. Композиция по п. 1, дополнительно содержащая по меньшей мере один агент, выбранный из ПЦР-буфера, иона соли, дНТФ, ДНК-полимеразы или нуклеиновой кислоты-мишени.
4. Набор для диагностики колоректального рака, содержащий композицию по любому из пп. 1-3.
5. Набор по п. 4, дополнительно содержащий по меньшей мере один из следующих реагентов:
реагент для экстракции ДНК, реагент для конверсии метилирования ДНК и реагент для очистки нуклеиновой кислоты.
6. Способ для выявления статуса метилирования генов в образце, в котором гены представляют собой ген ADHFE1, ген PPP2R5C и ген SDC2,
причем способ включает:
(1) экстрагирование геномной ДНК из биологического образца;
(2) получение продукта конверсии путем осуществления конверсии метилирования геномной ДНК, причем метилированный участок отличается от неметилированного участка вследствие обработки геномной ДНК метилированием; и
(3) осуществление с помощью реагентов флуоресцентного количественного ПЦР-выявления продукта конверсии для выявления статуса метилирования генов в образце;
(4) вычисление значения разницы между амплифицированными значениями Ct генов ADHFE1, PPP2R5C и SDC2 и амплифицированным значением Ct внутреннего эталонного гена GAPDH, и оценку степени метилирования гена образца на основе порога оценки, установленного на основе анализа клинических образцов известной патологической информации;
при этом:
реагент для выявления гена ADHFE1 содержит последовательности, представленные в SEQ ID NO: 7, SEQ ID NO: 8 и SEQ ID NO: 127;
реагент для выявления гена PPP2R5C содержит последовательности, представленные в SEQ ID NO: 53, SEQ ID NO: 54 и SEQ ID NO: 139; и
реагент для выявления гена SDC2 содержит последовательности, представленные в SEQ ID NO: 89, SEQ ID NO: 90 и SEQ ID NO: 101.
7. Способ по п. 6, в котором образец содержит по меньшей мере один образец, выбранный из образца ткани, образца крови, секрета или экскрементов.
8. Способ по п. 7, в котором экскременты содержат образец фекалий.
9. Способ по п. 6, в котором флуоресцентное количественное ПЦР-выявление осуществляли при 95°С в течение 10 минут, при 95°С в течение 15 секунд, при 55°С в течение 30 секунд и при 72°С в течение 30 секунд, в общем для 45 циклов.
10. Способ для оценивания болезненного состояния колоректального рака у субъекта для диагностики колоректального рака, включающий:
а) получение образца субъекта, где образец содержит нуклеиновые кислоты-мишени субъекта, а нуклеиновые кислоты-мишени представляют собой нуклеиновые кислоты гена ADHFE1, гена PPP2R5C и гена SDC2;
б) оценивание статуса метилирования нуклеиновых кислот-мишеней; и
в) оценивание болезненного состояния колоректального рака у субъекта основанное на статусе метилирования нуклеиновых кислот-мишеней;
при этом способ дополнительно включает: оценку болезненного состояния колоректального рака у субъекта с помощью флуоресцентного количественного метода ПЦР с использованием GAPDH в качестве внутреннего эталонного гена; определение, когда значение Ct GAPDH превышает 37, неудовлетворительного контроля качества; определение, когда значения Ct генов ADHFE1, PPP2R5C и SDC2, болезненного состояния колоректального рака у субъекта в виде отрицательного; и вычисление, когда значения Ct генов ADHFE1, PPP2R5C и SDC2 ≤ 37, разницы ΔCt между амплифицированными значениями Ct генов ADHFE1, PPP2R5C и SDC2 и амплифицированным значением Ct внутреннего эталонного гена GAPDH, и оценку болезненного состояния колоректального рака у субъекта на основе результатов кривой ROC и результатов AUC ΔCt генов ADHFE1, PPP2R5C и SDC2; когда ΔCt(ADHFE1) ≤ 8, ΔCt(PPP2R5C) ≤ 5 и ΔCt(SDC2) ≤ 12, болезненного состояния колоректального рака у субъекта в виде положительного;
в котором статус метилирования нуклеиновых кислот-мишеней оценивали на шаге б) с помощью реагентов,
при этом
реагент для выявления гена ADHFE1 содержит последовательности, представленные в SEQ ID NO: 7, SEQ ID NO: 8 и SEQ ID NO: 127;
реагент для выявления гена PPP2R5C содержит последовательности, представленные в SEQ ID NO: 53, SEQ ID NO: 54 и SEQ ID NO: 139; и
реагент для выявления гена SDC2 содержит последовательности, представленные в SEQ ID NO: 89, SEQ ID NO: 90 и SEQ ID NO: 101.
11. Способ по п. 10, в котором субъект представляет собой млекопитающее.
12. Способ по п. 10, дополнительно включающий:
выделение нуклеиновых кислот-мишеней из образца.
13. Способ по п. 10, дополнительно включающий:
амплификацию нуклеиновых кислот-мишеней.
| CN 111534590 A, 14.08.2020 | |||
| Fan J | |||
| et al | |||
| Genome-wide DNA methylation profiles of low-and high-grade adenoma reveals potential biomarkers for early detection of colorectal carcinoma.Clinical epigenetics, 2020, v | |||
| Способ гальванического снятия позолоты с серебряных изделий без заметного изменения их формы | 1923 |
|
SU12A1 |
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Паровой котел из трубчатых спиральной формы водяных элементов и с принудительной циркуляцией | 1928 |
|
SU22668A1 |
| СПОСОБ ДИАГНОСТИКИ ОНКОЛОГИЧЕСКИХ ЗАБОЛЕВАНИЙ | 2004 |
|
RU2245551C1 |
Авторы
Даты
2025-03-10—Публикация
2021-04-23—Подача