Перекрестная ссылка на родственные заявки
Настоящая заявка испрашивает приоритет в соответствии с разделом 119(e) предварительной заявки на патент США No.62/774,994, поданной 4 декабря 2018 г., с названием «DNA METHYLATION MARKERS FOR EARLY DETECTION OF CERVICAL CANCER», содержание которой включено в настоящее изобретение путём ссылки.
Перечень последовательностей
Настоящая заявка содержит Перечень последовательностей, который был подан в электронном виде в формате ASCII и полностью включен в настоящее изобретение путём ссылки. Указанная копия ASCII, созданная 12 января 2020 г., называется TPC53811 Seq List_ST25.txt и имеет размер 36 864 байта.
Область техники, к которой относится изобретение
Настоящее изобретение в основном относится к сигнатурам метилирования ДНК в ДНК человека, особенно в области молекулярной диагностики. Более конкретно, настоящее изобретение относится к биомаркерам метилирования ДНК в виде панели, индивидуальных, а также комбинации полигенных биомаркеров метилирования ДНК для раннего выявления, а также скрининга рака шейки матки, и их применению в качестве диагностического набора для раннего и точного выявления рака шейки матки.
Уровень техники изобретения
Рак остается основной причиной смерти у людей. Раннее выявление рака может значительно улучшить показатели излечения и снизить огромные личные и финансовые затраты пациентов, их семей и системы здравоохранения. В то же время, скрининг здоровых людей для оценки экспрессии биомаркеров на предраковой стадии и их изменений применим в методологии скрининга в масштабах всей популяции и помогает выявлять здоровых людей, подверженных риску развития рака, восприимчивых к раку. Рак шейки матки не является исключением. Скрининг может выявить рак на ранней стадии - до того, как он вызовет симптомы. Если рак шейки матки обнаружен на самой ранней стадии, шанс выживания составляет примерно 93%, а на последних стадиях он снижается до 15% https://www.cancer.org/cancer/cervical-cancer/detection-diagnosis-staging/survival.html. Современные методы скрининга включают мазки по Папаниколау, жидкостную цитологию, тестирование на ВПЧ и визуальный осмотр, однако надежный высокоточный и чувствительный способ раннего выявления рака шейки матки отсутствует.
Биомаркеры составляют одну из важнейших областей диагностики рака. Биомаркеры рака особенно полезны для раннего выявления или диагностики заболевания. Биомаркеры можно использовать для скрининга пациентов, для классификации различных стадий или степеней рака и для составления прогноза и прогнозирования устойчивости к терапии.
Общепризнанное открытие того, что вирус папилломы человека (ВПЧ) является возбудителем неоплазии шейки матки, произвело революцию в методах предотвращения и лечения этого гинекологического заболевания с вспомогательных позиций (молекулярное тестирование на ВПЧ) (1). Знание генотипа ВПЧ действительно полезно для клинического прогнозирования, поскольку ВПЧ типов 16 и/или 18 связан с большим риском прогрессирования поражения, чем другие канцерогенные типы. Однако стойкая инфекция канцерогенными генотипами ВПЧ является необходимым предшественником и движущей силой канцерогенеза шейки матки. Последний представляет собой поэтапное прогрессирование от предраковых стадий (дисплазия шейки матки, ДШМ) к инвазивному раку шейки матки. ДШМ низкой степени (ДШМ1) в высшей степени обратима, тогда как ДШМ высокой степени 2 и 3 (то есть ДШМ2 и ДШМ3, соответственно) имеет высокий риск прогрессирования инвазии, то есть рака шейки матки. Это особенно верно для ДШМ3.
Ведение женщин с патологиями ДШМ в клинике по-прежнему представляет собой серьезную дилемму для гинекологов, поскольку агрессивное абляционное или эксцизионное лечение может вызвать немедленные осложнения или повысить риск выкидыша или преждевременных родов в более позднем возрасте, когда пациентка решит забеременеть. Недавние данные свидетельствуют о том, что эпигенетические изменения в определенных генах могут опосредовать или предсказать канцерогенное прогрессирование. Биомаркер раннего выявления рака может однозначно дифференцировать редкие клетки с поражениями на бессимптомной и предраковой стадиях, благодаря значительным изменениям, которые включают в себя биохимические изменения на эпигенетическом уровне. Эти эпигенетические изменения в качестве биомаркеров довольно часто вырабатываются в аномально больших количествах в раковых тканях и часто препятствуют проявлению самого заболевания. Для выявления молекулярных изменений, происходящих задолго до начала и прогрессирования заболевания, чрезвычайно важна разработка молекулярных биомаркеров. Было показано, что уровни одного из таких эпигенетических биомаркеров - уровни метилирования ДНК определенных сайтов CpG в вирусных генах и генах хозяина - увеличиваются с тяжестью первичных поражений шейки матки (2-7).
К числу наиболее изученных и целевых генов хозяина с эпигенетическими изменениями, связанными с раком шейки матки и его предшественниками, относятся молекула клеточной адгезии 1 (CADM1); ассоциированная со смертью протеинкиназа 1 (DAPK1); миелин и лимфоциты, белок дифференцировки Т-клеток (MAL); парный бокс 1 (PAX1); теломеразная обратная транскриптаза (TERT); белок 3, подобный полосе 4.1 белка мембраны эритроцитов (EPB41L3), член 1 семейства доменов ассоциации Ras (RASSF1); SRY-box 1 (SOX1); кадгерин 1 (CDH1); LIM гомеобоксный фактор транскрипции 1 альфа (LMX); циклин A1 (CCNA1); семейство со сходством последовательностей из 19 членов A4, хемокиноподобный мотив C-C (FAM19A4); и рецептор ретиноевой кислоты бета (RARβ)8. Были исследованы одиночные (9) маркеры метилирования в дополнение к тем, которые включали два (т.е., CADM1 и MAL (3,4,10); MAL и miR124-2 (11-14), три (т.е., CADM1, MAL и miR124-2) (13,15), четыре (т.е., JAM3, EPB41L3, TERT и C13ORF18) (16,17) и пять (т.е., PAX1, DAPK1, RARβ, WIF1 и SLIT2) (14) маркеров в панели, а также панели, включающие различные комбинации маркеров SOX1, PAX1, LMX1A и NKX6-1 для достижения достаточно высокой чувствительности для запущенных поражений (18).
Однако только одно предыдущее исследование с использованием подхода изучения метилирования по всему геному выявило три панели метилирования (JAM3/ANKRD18CP, C13ORF18/JAM3/ANKRD18CP и JAM3/GFRA1/ANKRD18CP) с самой высокой комбинированной диагностической точностью для обнаружения ДШМ2+ в образцах шейки матки; и их чувствительность составила 72%, 74% и 73%, соответственно, с соответствующей специфичностью 79%, 76% и 77% (2). Соответственно, существует потребность в улучшенных способах идентификации биомаркеров метилирования ДНК, панели биомаркеров метилирования ДНК, связанных с ранним выявлением и прогнозированием риска рака шейки матки, а также в наборах реагентов, основанных на таких биомаркерах, для скрининга в масштабах популяции клинически здоровых женщин с целью раннего выявления рака шейки матки и предрасположенности к нему, а также для оценки риска его развития у женщин с предраковыми патологиями.
Настоящее изобретение относится к решению проблемы, связанной с отсутствием маркеров раннего выявления рака шейки матки, путём использования биомаркеров метилирования ДНК в качестве единичных, комбинированных, а также панельных биомаркеров, поскольку отсутствует один или комбинированный маркер метилирования, который обладает соответствующими диагностическими характеристиками для прогнозирования риска рака шейки матки на ранней стадии в настоящее время. Настоящее изобретение относится к способу получения ранних биомаркеров прогрессирования предракового поражения до рака шейки матки, которые можно использовать для общего скрининга у женщин без симптомов, а также у женщин с патологиями от ДШМ1 до ДШМ3.
Задачи изобретения
Основная задача настоящего изобретения относится к биомаркерам для раннего выявления и диагностики рака шейки матки у человека.
Другая задача настоящего изобретения относится к раскрытому здесь in vitro способу, называемому «анализ прогрессирующих изменений метилирования ДНК (APDMA)», который включает в себя этапы изучения полногеномных профилей метилирования ДНК образцов, полученных от женщин с различными степенями патологии ДШМ (от ДШМ1 до ДШМ3), по сравнению с контрольными образцами, полученными от здоровых женщин, для получения CG-идентификаторов (CGID) в качестве биомаркеров метилирования ДНК, которые при объединении предсказывают, используя описанную здесь модель линейной регрессии, рак шейки матки с чувствительностью и специфичностью > 95% в общедоступных профилях метилирования рака шейки матки.
Другая задача настоящего изобретения относится к молекулярным биомаркерам в качестве индикаторов популяционного скрининга женщин для раннего выявления рака шейки матки, а также для оценки риска развития рака шейки матки у женщин с патологиями от ДШМ1 до ДШМ3.
Еще одна задача настоящего изобретения относится к чипу/матрице, пригодному для раннего выявления и диагностики рака шейки матки.
Еще одна задача настоящего изобретения относится к более дешевому, точному, надежному, высокочувствительному, специфичному и высокопроизводительному диагностическому набору для точной ранней диагностики рака шейки матки человека, пригодному для применения любым специалистом в данной области техники.
Сущность изобретения
Соответственно, настоящее изобретение относится к способам и материалам, пригодным для изучения изменений метилирования ДНК, и относится к CGID биомаркерам метилирования ДНК для раннего выявления и диагностики рака шейки матки, при котором прогрессирование предраковых поражений шейки матки (дисплазия шейки матки, ДШМ 1-3 степени) коррелирует с повышенной частотой метилирования ДНК в положениях CG в геноме человека, в форме зонда-идентификатора Illumina или числа метилирования ДНК или CG-идентификаторов (CGID), которые получают с использованием раскрытого в настоящем изобретении in vitro способа «анализа прогрессирующих изменений метилирования ДНК» (APDMA), как описано в настоящем изобретении. Как подробно обсуждается ниже, обычно эти биомаркеры основаны на переменных, которые позволяют прогнозировать риск у женщин с патологиями от ДШМ1 до ДШМ3, а также в популяционных скринингах на развитие рака шейки матки и, в свою очередь, пригодны в качестве биомаркеров раннего выявления и диагностики. Настоящее изобретение предусматривает, что указанные положения биомаркеров CGID почти однородно метилированы при раке шейки матки и почти однородно неметилированы в образцах из нормальной шейки матки. Таким образом, настоящее изобретение относится к указанному набору «однозначно» различных профилей метилирования ДНК, которые создают бинарную дифференциацию между раком шейки матки и доброкачественными тканями по профилю метилирования ДНК в этих сайтах CGID, в результате чего эти сайты метилированы только при раке шейки матки и полностью неметилирован в незлокачественной ткани. Более того, как раскрыто в настоящем изобретении, эти сайты биомаркеров показывают возрастающую частоту метилирования ДНК по мере прогрессирования предраковых поражений шейки матки, переходящих от ДШМ1 к ДШМ3. Таким образом, настоящее изобретение относится к способу раннего выявления и диагностики in vitro с использованием направленной амплификации указанных биомаркеров CGID и глубокого бисульфитного секвенирования следующего поколения для обнаружения даже нескольких молекул клеток рака шейки матки или даже клеток из предраковых поражений на пути к превращению в рак шейки матки на фоне преимущественно нормального клеточного профиля шейки матки. Таким образом, настоящее изобретение применимо для пока еще недоступного раннего обнаружения клеток рака шейки матки на высоком фоне незлокачественной ткани, в частности, с использованием цервикальных образцов, таких как мазки по Папаниколау, в качестве простого и удобного метода раннего обнаружения, пригодного для использования любым специалистом в данной области техники.
Вариант осуществления настоящего изобретения относится к in vitro способу получения высокопрогнозируемых сайтов для рака шейки матки для его раннего выявления даже на бессимптомных и предраковых стадиях, называемому «способ анализа прогрессирующих изменений метилирования ДНК (APDMA)», с использованием различных источников данных о метилировании ДНК по всему геному, полученных с помощью секвенирования следующего поколения, включая чипы MeDIP, секвенирование MeDIP и т.д., полученные в виде сигнатур CGID биомаркеров метилирования ДНК. Настоящее изобретение относится к комбинации «однозначных» CGID биомаркеров для выявления рака шейки матки в исследуемом наборе данных со всего генома, полученных из образцов прогрессирующих предраковых поражений, переходящих от ДШМ1 к ДШМ3.
Предыдущие способы анализы, разработанные до настоящего изобретения, в которых использовался классический дизайн «случай-контроль» и логистическая регрессия, выявили CGID биомаркеры метилирования ДНК, которые обнаруживают рак с более низкой чувствительностью и специфичностью. Таким образом, другой вариант осуществления настоящего изобретения относится к реализованному на компьютере способу получения кандидатных биомаркеров метилирования ДНК для раннего выявления для диагностики рака шейки матки, называемому способ APDMA, который выявляет самые ранние профили метилирования рака, которые являются первичными и важными для ракового состояния и, таким образом, присутствуют во всех образцах рака шейки матки, протестированных при создании настоящего изобретения.
Вариант осуществления настоящего изобретения относится к in vitro способу, который точно определяет рак шейки матки путём измерения метилирования ДНК в полигенном наборе CGID биомаркеров у сотен людей одновременно, путём последовательной амплификации с праймерами, специфичными для мишени, затем с праймерами, специфичными для «штрих-кодов» и мультиплексного секвенирования в одной реакции секвенирования нового поколения Miseq, с последующим извлечением данных и количественной оценки метилирования.
Вариант осуществления настоящего изобретения относится к способу измерения метилирования указанных CGID биомаркеров метилирования ДНК in vitro с использованием метода пиросеквенирования или ПЦР, специфичной для метилирования. В настоящем изобретении раскрыт расчет полигенного взвешенного показателя метилирования, который позволяет прогнозировать рак шейки матки.
Вариант осуществления настоящего изобретения относится к панели биомаркеров метилирования ДНК для скрининга, диагностики, раннего выявления и прогнозирования рака шейки матки в образце ДНК, выделенной из образца, взятого у женщины, включая женщин, не имеющих других клинических признаков рака шейки матки, из образцов шейки матки.
Вариант осуществления настоящего изобретения относится к панели биомаркеров метилирования ДНК в форме чипа для скрининга, диагностики, раннего выявления и прогнозирования рака шейки матки в образце ДНК, выделенной из образца, взятого у женщины, включая женщин, не имеющих других клинических признаков рака шейки матки, из образцов шейки матки.
Вариант осуществления настоящего изобретения относится к неинвазивному способу in vitro с использованием панели биомаркеров метилирования ДНК для скрининга, диагностики, раннего выявления и прогнозирования рака шейки матки в образце ДНК, выделенной из образца, взятого у женщины, включая женщин, не имеющих других клинических признаков рака шейки матки, из образцов шейки матки.
Вариант осуществления настоящего изобретения относится к применению описанных здесь биомаркеров метилирования ДНК для скрининга, диагностики, раннего выявления и прогнозирования рака шейки матки в образце ДНК, выделенной из образца, взятого у женщины, включая женщин, не имеющих других клинических признаков рака шейки матки, из образцов шейки матки.
Настоящее изобретение относится к надежным биомаркерам метилирования ДНК, идентифицированным с использованием положений CGID в геноме человека, которые обеспечивают высокоточную, специфичную и чувствительную оценку риска, которая может определять раннее вмешательство и лечение рака шейки матки даже у женщин на бессимптомных и предраковых стадиях. Настоящее изобретение обеспечивает простой, но эффективноый способ, который может быть применен любым специалистом в данной области техники для обнаружения рака шейки матки. Настоящее изобретение относится к применению описанных здесь CGID биомаркеров метилирования ДНК для популяционного скрининга здоровых женщин на выявление у них рака шейки матки, а также для мониторинга и оценки риска рака у женщин с инфекцией ВПЧ и предраковыми поражениями ДШМ. Настоящее изобретение демонстрирует применимость раскрытых биомаркеров метилирования ДНК при выявлении рака шейки матки в образцах ДШМ с использованием полигенной оценки на основе описанных здесь способов измерения метилирования ДНК. Настоящее изобретение также раскрывает применимость раскрытого способа получения «полигенных» CGID биомаркеров метилирования ДНК для выявления рака шейки матки с использованием любого метода, доступного специалистам в данной области техники для полногеномного бисульфитного секвенирования, такого как бисульфитное секвенирование следующего поколения, секвенирование MeDip, ионное торрент-секвенирование, чипы Illumina 450 K и микрочипы Epic и т.д., с последующим применением описанного здесь способа APDMA для обнаружения специфичных и чувствительных маркеров, пригодных для раннего и очень раннего выявления рака шейки матки, благодаря наличию у них однозначных различий в профилях метилирования ДНК между контрольными образцами здоровых людей и образцами рака шейки матки с постепенным увеличением частоты при переходе от образцов от предраковых стадий ДШМ1 к ДШМ3.
Другие задачи, признаки и преимущества настоящего изобретения станут очевидны специалистам в данной области техники из следующего подробного описания. Однако следует понимать, что подробное описание и конкретные примеры, указывающие на некоторые варианты осуществления настоящего изобретения, даны в качестве иллюстрации, а не ограничения. Многие изменения и модификации в пределах объема настоящего изобретения могут быть сделаны без отступления от его сущности, и настоящее изобретение включает в себя все такие модификации.
Краткое описание чертежей
Фигура 1. Блок-схема для разработки способа анализа прогрессирующих изменений метилирования ДНК (APDMA) для получения биомаркеров метилирования ДНК раннего выявления. Блок-схема описывает аналитическую процедуру для разработки способа APDMA на основе профиля метилирования ДНК с использованием идентификационного анализа с зондом Illumina (CGID), который однозначно дифференцирует нормальный профиль образцов шейки матки от профиля метилирования ДНК в образцах рака шейки матки для получения «однозначных» CGID биомаркеров метилирования ДНК для раннего выявления, диагностики, а также скрининга на рак шейки матки. На этапе 1 измерения метилирования ДНК проводят у образцов шейки матки с предраковыми поражениями на стадиях от ДШМ1 до ДШМ3 в сравнении со здоровыми контрольными образцами, указанные измерения метилирования ДНК получают либо путём проведения анализа выделенной из образцов ДНК на чипе Illumina Beadchip 450K или 850K, либо путём проведения пиросеквенирования выделенной из образца ДНК, либо с помощью масс-спектрометрии (Epityper™), либо с помощью анализа метилирования на основе ПЦР и целевой амплификации области, охватывающей целевые CGID, описанные здесь, из преобразованной бисульфитом ДНК, с последующим штрих-кодированием во втором раунде амплификации и индексированного мультиплексного секвенирования на секвенаторе нового поколения Illumina. На этапе 2 выполняется способ статистического анализа для измерений метилирования ДНК, сделанных на этапе 1, где статистический анализ включает в себя анализ соотношений правильного и ложного обнаружения сигналов (ROC), анализ иерархической кластеризации или анализ нейронной сети. На этапе 3 осуществляется разработанный и раскрытый в настоящем изобретении способ «анализа прогрессирующих изменений метилирования ДНК» (APDMA) для определения положений CGID, уровни метилирования которых являются ранним предиктором или биомаркером рака шейки матки. На этапе 4 настоящее изобретение предусматривает дополнительное сужение и сокращение списка комбинаций метилирования ДНК полигенных CGID до набора биомаркеров из 16 CGID. Этот способ позволяет получить «однозначные», а не количественные различия в профилях метилирования между нормальными клетками и клетками рака шейки матки, что, в свою очередь, позволяет проводить раннее выявление, благодаря характерному переключению профиля метилирования ДНК в выбранных CGID, которые служат в качестве биомаркеров метилирования ДНК для раннего выявления, диагностики и скрининга рака шейки матки. Они служат в качестве панели кандидатов на CGID биомаркеры для раннего выявления рака шейки матки у женщин, особенно тех, у которых нет симптомов или есть предраковые поражения.
Фигура 2. Способ получения сайтов, частота метилирования которых прогрессивно увеличивается у предраковых стадий ДШМ. ДНК, полученная из образцов шейки матки из гистологических образцов пациентов с ДШМ1, ДШМ2 и ДШМ3; и нетрансформированные контрольные образцы от здоровых людей подвергали полногеномному анализу метилирования ДНК на чипах Illumina Epic. Уровень метилирования 7715 CGID достоверно коррелировал (q> 0,05) с прогрессированием предраковых стадий ДШМ от ДШМ1 к ДШМ3. A. Представление в браузере IGV различий в метилировании этих сайтов по всему геному по сравнению с контрольными образцами шейки матки. Верхний трек показывает положение хромосом. Второй трек показывает положение референсных последовательностей генов в геноме. Следующие треки (ΔCIN1-Ctrl, ΔCIN2-Ctrl, ΔCIN3-Ctrl) показывают разницу в среднем уровне метилирования между каждой из стадий ДШМ и контролем. Наблюдается прогрессирующее стадийное гиперметилирование.
Фигура 3. Сайты, полученные с помощью способа APDMA, однозначно различаются у образцов нормальной шейки матки и образцов рака шейки матки. A. Карта интенсивности показывает, что 79 главных CGID, частота метилирования которых увеличивается во время прогрессирования предраковых стадий шейки матки, выявляют рак шейки матки с использованием данных о метилировании ДНК у 270 пациентов (GSE68339). CGID демонстрируют совершенно различные профили метилирования при раке шейки матки и у нормальной шейки матки. Они полностью неметилированы в нормальной ткани и сильно метилированы в раковой ткани. B. Среднее метилирование для каждой из групп нормальных тканей, предраковых стадий и рака шейки матки (от ДШМ1 до ДШМ3) (синий цвет означает 0% метилирования, а темно-красный означает 100% метилирования).
Фигура 4. Специфичность и специфичность показателя метилирования бигенной ДНК, полученного с использованием способа APDMA для выявления ДНК рака шейки матки в независимой когорте. A. Расчет величины эффекта, регрессия со штрафом и многомерная линейная регрессия кратко определили подмножество двух CGID, и было вычислено уравнение линейной регрессии для прогнозирования рака шейки матки. B. Порог обнаружения рака был рассчитан при помощи ROC. C. Используя этот порог, чувствительность и специфичность этого комбинированного набора маркеров равна 1, а AUC равна 1.
Фигура 5. Показатель метилирования рака в отдельных образцах от контрольных ДШМ1 до ДШМ3 и пациентов с раком шейки матки. A. Показатели метилирования (прогноз рака шейки матки), рассчитанные с использованием уравнения, представленного на Фигуре 4A, для каждого из отдельных образцов из контрольной группы, от ДШМ1 до ДШМ3 и рака шейки матки, демонстрирующие увеличение показателей метилирования при прогрессирующих предраковых поражениях. B. График разброса, показывающий средний показатель метилирования для контрольной, предраковой и раковой групп.
Фигура 6. Корреляция между оценкой бигенного метилирования и прогрессированием от контроля через предраковые стадии до рака шейки матки. Образцы рака шейки матки от GSE68339 ДШМ1 до ДШМ3 взяты из когорты Мак-Гилла, описанной в этой заявке (присвоенный ранг Спирмена: контроль: 0, от ДШМ1 до ДШМ3: 1-3, рак шейки матки: 4).
Фигура 7. Валидация маркера метилирования рака шейки матки с использованием данных о метилировании ДНК из TCGA (Атлас ракового генома) (n = 312). Поскольку данные для рака шейки матки были доступны в TCGA только для одного CGID (cg13944175), авторы настоящего изобретения провели оценку метилирования для рака шейки матки, используя уравнение линейной регрессии с данными метилирования ДНК только для CGID cg13944175. Корреляцию Пирсона рассчитывали между стадией прогрессирования рака и показателем метилирования (см. Статистику в A и диаграмму корреляции в B). ДШМ1 - ДШМ 3 относятся к когорте Мак-Гилла, описанной в этой заявке. Присвоенные шкалы: контроль: 0, от ДШМ1 до ДШМ3: 1-3, рак шейки матки: 4.
Фигура 8. Применение настоящего изобретения: Прогнозирование рака шейки матки в образцах от ДШМ1 до ДШМ3. Не у всех пациентов с ДШМ-1-3 развивается рак шейки матки, хотя его доля у пациентов с ДШМ3 выше, чем у пациентов с ДШМ1. В настоящем изобретении проверяется, можно ли использовать показатель метилирования, представленный на Фигуре 3, для идентификации отдельных пациентов, которые имеют показатель метилирования рака шейки матки, как доказательство применимости настоящего изобретения. A. По оси X нанесены отдельные пациенты, группы обозначены линиями под осью X. Ось Y показывает прогноз развития рака (1) и отсутствие причин рака (0). B. Количество людей с прогнозом рака в каждой группе. Как и ожидалось, прогнозирование рака увеличивается с ДШМ1 до ДШМ3.
Подробное описание изобретения
В описании вариантов осуществления изобретения может быть сделана ссылка на прилагаемые чертежи, которые составляют его часть и на которых в качестве иллюстрации показан конкретный вариант осуществления изобретения, в котором изобретение может быть реализовано на практике. Следует понимать, что могут использоваться другие варианты осуществления изобретения и могут быть внесены структурные изменения, не выходящие за рамки объема настоящего изобретения. Многие из описанных или упомянутых здесь методик и процедур хорошо известны и обычно используются специалистами в данной области техники. Если не указано иное, то все термины из данной области техники, обозначения и другие научные термины или терминология, используемые в данном описании, имеют значения, обычно понятные специалистам в данной области, к которой относится настоящее изобретение. В некоторых случаях термины с общепринятыми значениями определены здесь для ясности и/или для удобства ссылок, и включение таких определений в настоящее изобретение не обязательно должно толковаться как представляющее существенное отличие от того, что обычно понимается в данной области техники.
Все иллюстрации чертежей предназначены для описания выбранных вариантов настоящего изобретения и не предназначены для ограничения объема настоящего изобретения.
Все упомянутые здесь публикации включены в настоящее изобретение путём ссылки для раскрытия и описания аспектов, способов и/или материалов в связи с процитированными публикациями.
Метилирование ДНК относится к химическим модификациям молекулы ДНК. Было обнаружено, что технологические платформы, такие как микрочип Illumina Infinium или способы, основанные на секвенировании ДНК, позволяют проводить высоконадежные и воспроизводимые измерения уровней метилирования ДНК человека. В геноме человека более 28 миллионов локусов CpG. Следовательно, определенным локусам присваиваются уникальные идентификаторы, такие как те, что находятся в базе данных локусов CpG Illumina (см., например, Technical Note: Epigenetics, CpG Loci Identification ILLUMINA Inc. 2010). Эти идентификаторы обозначения локуса CG используются в настоящем изобретении.
Определения:
Используемый здесь термин «CG» или «CpG», используется взаимозаменяемо и относится к динуклеотидной последовательности в ДНК, содержащей основания цитозина и гуанозина. Эти динуклеотидные последовательности могут метилироваться в ДНК человека, а также других животных. CGID раскрывает свое положение в геноме человека, как это определено манифестом Illumina 450K или манифестом Illumina EPIC (аннотации перечисленных здесь CG общедоступны по адресу https://bioconductor.org/packages/release/data/annotation/html/ IlluminaHumanMethylation450k.db.html или https://bioconductor.org/packages/release/data/annotation/html/IlluminaHumanMethylationEPICmanifest.html и установлены как пакет R IlluminaHumanMethylation450k.db (пакет R версии 2.0.9.) или IlluminaHumanMethylationEPICmanifest (пакет R версия 0.3.0).
Используемый здесь термин «бета-значение» относится к вычислению уровня метилирования в положении CGID, полученного путём нормализации и количественной оценки чипов Illumina 450K или EPIC с использованием отношения интенсивностей между метилированными и неметилированными зондами и формулы: значение бета = Интенсивность метилированного C / (интенсивность метилированного C + интенсивность неметилированного C) от 0 до 1, где 0 - полностью неметилированный, а 1 - полностью метилированный.
Используемый здесь термин «регрессия со штрафом» относится к статистическому методу, направленному на выявление наименьшего количества предикторов, необходимых для прогнозирования результата из большего списка биомаркеров, как реализовано, например, в статистическом пакете R, «штрафуемому», как описано в Goeman, J. J., L1 penalized estimation in the Cox proportional hazards model. Biometrical Journal 52(1), 70-84.
Используемый здесь термин «кластеризация» относится к группировке набора объектов таким образом, что объекты в одной группе (называемой кластером) более похожи (в том или ином смысле) друг на друга, чем на объекты в других группах (кластерах).
Используемый здесь термин «иерархическая кластеризация» относится к статистическому методу, который строит иерархию «кластеров» на основе того, насколько похожи (близки) или несходны (отдалены) кластеры друг от друга, как описано, например, в Kaufman, L.; Rousseeuw, P.J. (1990). Finding Groups in Data: An Introduction to Cluster Analysis (1 ed.). New York: John Wiley. ISBN 0-471-87876-6.
Используемый здесь термин «анализ соотношений правильного и ложного обнаружения сигналов (ROC)» относится к статистическому методу, который создает график, который иллюстрирует работу предиктора. Истинно-положительный показатель прогноза наносится на график против ложноположительного показателя при различных настройках порогового значения для предиктора (т.е. разный % метилирования), как описано, например, в Hanley, James A.; McNeil, Barbara J. (1982). «The Meaning and Use of the Area under a Receiver Operating Characteristic (ROC) Curve». Radiology 143 (1): 29-36.
Используемый здесь термин «многомерная или полигенная линейная регрессия» относится к статистическому методу, который оценивает взаимосвязь между несколькими «независимыми переменными» или «предикторами», такими как процент метилирования в нескольких CGID, и «зависимой переменной», такой как рак. Этот метод определяет «вес» или коэффициент каждого CGID при прогнозировании «результата» (зависимой переменной, такой как рак), когда в модель включены несколько «независимых переменных», таких как CGID.
Используемый здесь термин «эпигенетический» означает относящийся к химической модификации молекулы ДНК, являющийся или включающий в себя химическую модификацию молекулы ДНК. Эпигенетические факторы включают в себя добавление или удаление метильной группы, что приводит к изменению уровней метилирования ДНК. Новые молекулярные биомаркеры раннего выявления, диагностики или прогнозирования рака шейки матки, которые основаны на выявлении паттернов метилирования в геномной ДНК, такие как те, которые описаны в настоящем изобретении как биомаркеры на основе CGID, позволяют прогнозировать риск и предрасположенность к раку шейки матки даже на очень ранних стадиях, когда у женщин отсутствуют симптомы или на предраковых стадиях, прогрессирующих от ДШМ1 до ДШМ3, и они могут применяться в клинической практике эпидемиологами, медицинскими работниками, и раскрытие настоящего изобретения сделано таким образом, чтобы оно было доступным и пригодным для применения любым специалистом в данной области техники. Исключительно клинические биомаркеры, такие как мазки по Папаниколау, гистологическая идентификация, имеют долгую и успешную историю в диагностике рака шейки матки, однако их недостатком является большая степень вариабельности, и они не могут использоваться для раннего выявления рака шейки матки. Напротив, молекулярные биомаркеры, такие как эпигенетические маркеры в форме биомаркеров метилирования ДНК, до сих пор применяются редко.
Используемый здесь термин «биомаркер метилирования ДНК» относится к положению CpG, которое потенциально метилировано. Метилирование обычно происходит в CpG-содержащей нуклеиновой кислоте. CpG-содержащая нуклеиновая кислота может присутствовать, например, в CpG-островке, дублете CpG, промоторе, интроне или экзоне гена. Например, в предоставленных здесь генетических областях потенциальные сайты метилирования охватывают промоторные/энхансерные области указанных генов. Таким образом, области могут начинаться до промотора гена и распространяться дальше в транскрибируемую область.
В соответствии со способом настоящего изобретения предполагается, что частота клеток, которые демонстрируют профиль метилирования ДНК рака шейки матки, увеличивается с прогрессированием от патологий ДШМ1 к ДШМ3, и что эти профили метилирования характерны для самого раннего рака шейки матки. Во-вторых, поскольку клетки, которые превращаются в рак, редко встречаются на ранних стадиях предракового развития, профиль метилирования ДНК должен однозначно отличаться от нормального профиля клеток шейки матки, чтобы его можно было обнаружить на фоне в основном незлокачественных клеток на самых ранних стадиях. В-третьих, эти профили метилирования ДНК должны присутствовать во всех образцах полностью развитого рака шейки матки, если они являются первичной и критической характеристикой рака шейки матки. Принимая во внимание три вышеупомянутых предварительных условия, способ in vitro настоящего изобретения, называемый «анализ прогрессирующих изменений метилирования ДНК (APDMA)», включает в себя этапы изучения полногеномных профилей метилирования ДНК образцов, выделенных и полученных от женщин с различными стадиями патологии ДШМ (от ДШМ1 до ДШМ3) по сравнению со здоровыми, нетрансформированными, здоровыми контрольными образцами шейки матки после хорошо охарактеризованного генотипирования ВПЧ с использованием чипов Infinium Methylation EPIC. Настоящее изобретение относится к способу in vitro для получения зонда-идентификатора Illumina, числа метилирования ДНК или CG-идентификаторов (CGID) в качестве биомаркеров метилирования ДНК, которые при объединении предсказывают, используя описанную здесь модель линейной регрессии, рак шейки матки с чувствительностью и специфичностью > 95% в общедоступных профилях метилирования рака шейки матки. Настоящее изобретение также обеспечивает панель биомаркеров метилирования ДНК для скрининга и раннего выявления рака шейки матки, где панель включает в себя CGID, имеющие последовательности, выбранные из группы, состоящей из SEQ ID NO:1 - SEQ ID NO:79, как указано в Таблице 1, и их комбинации, такие как включенные в короткий список подмножества последовательностей Таблицы 1, перечисленные в Таблице 2, и более короткий список подмножества CGID, перечисленные в Таблице 3, соответственно, как раскрыто здесь ниже. Таким образом, настоящее изобретение относится к двум CGID, минимально достаточных для обнаружения рака шейки матки в общедоступных данных о метилировании ДНК с чувствительностью и специфичностью, приближающейся к 1. Настоящее изобретение также относится к наборам для измерения in vitro биомаркеров метилирования ДНК в качестве уровней метилирования ДНК раскрытых CGID в ДНК, выделенной из образцов шейки матки, которые будут использоваться для популяционного скрининга женщин для раннего выявления рака шейки матки, а также для оценки риска рака шейки матки у женщин с патологиями от ДШМ1 до ДШМ3.
Настоящее изобретение имеет несколько вариантов осуществления. В одном из вариантов осуществления настоящее изобретение относится к биомаркерам CGID полигенного метилирования ДНК рака шейки матки в мазках из шейки матки для раннего выявления рака шейки матки, указанная панель биомаркеров полигенного метилирования ДНК получена с использованием «способа анализа прогрессирующих изменений метилирования ДНК (APDMA)», раскрытого в настоящем изобретении, на основе данных о метилировании ДНК по всему геному, полученных с помощью методов картирования, таких как чипы Illumina 450K или 850K, полногеномного бисульфитного секвенирования с использованием различных платформ секвенирования следующего поколения, секвенирования методом иммунопреципитации метилированной ДНК (MeDIP) или гибридизации с чипами олигонуклеотидов.
В одном из вариантов осуществления настоящее изобретение относится к способу получения биомаркеров метилирования ДНК для выявления рака шейки матки, включающему в себя этап выполнения статистического анализа и способ «анализа прогрессирующих изменений метилирования ДНК (APDMA)», раскрытый в настоящем изобретении для измерения метилирования ДНК, полученной из образцов шейки матки предраковых поражений от ДШМ1 до ДШМ3.
В одном из вариантов осуществления изобретения раскрытый здесь способ включает в себя выполнение статистического анализа и способа «анализа прогрессирующих изменений метилирования ДНК (APDMA)» измерения метилирования ДНК, полученной из образцов шейки матки, указанное измерение метилирования ДНК получают путём выполнения анализа Illumina Beadchip 450K или 850K извлеченной из образцов ДНК. В другом варианте осуществления изобретения указанное измерение метилирования ДНК проводят путём выполнения пиросеквенирования ДНК, выделенной из образца, или с помощью масс-спектрометрии (Epityper™), или с помощью анализов метилирования на основе ПЦР и целевой амплификации области, охватывающей целевые CGID, описанные здесь, из преобразованной бисульфитом ДНК, с последующим штрих-кодированием во втором раунде амплификации и индексированного мультиплексного секвенирования на секвенаторе нового поколения Illumina. В дополнительном варианте осуществления изобретения указанный статистический анализ включает в себя анализ соотношений правильного и ложного обнаружения сигналов (ROC). В еще одном варианте осуществления изобретения указанный статистический анализ включает в себя анализ иерархической кластеризации. В дополнительном варианте осуществления изобретения указанный статистический анализ включает в себя нейросетевой анализ.
В одном из вариантов осуществления настоящее изобретение относится к in vitro способу получения ранних предикторов рака шейки матки, включающему в себя следующие этапы: (a) измерение метилирования образца ДНК, выделенного из образца шейки матки, (b) выполнение статистического анализа результатов измерения метилирования ДНК, полученных на этапе a, (c) определение статуса метилирования ДНК множества независимых геномных положений CG, называемых CG-идентификаторами (CGID), путём выполнения анализа прогрессивных изменений метилирования ДНК (APDMA) профилей метилирования ДНК всего генома, полученных на этапе b, (d) классификация CGID на основе частоты их метилирования ДНК, коррелирующей с прогрессированием предраковой стадии рака шейки матки, (e) получение кандидатных CGID из классификации на этапе d для получения ранних предикторов рака шейки матки в виде биомаркеров метилирования ДНК.
В другом варианте осуществления настоящее изобретение относится к in vitro способу получения ранних предикторов рака шейки матки, включающему в себя следующие этапы: (a) измерение метилирования образца ДНК, выделенного из образца шейки матки, (b) выполнение статистического анализа результатов измерения метилирования ДНК, полученных на этапе a, (c) определение статуса метилирования ДНК множества независимых геномных положений CG, называемых CG-идентификаторами (CGID), путём выполнения анализа прогрессивных изменений метилирования ДНК (APDMA) профилей метилирования ДНК всего генома, полученных на этапе b, (d) классификация CGID на основе частоты их метилирования ДНК, коррелирующей с прогрессированием предраковой стадии рака шейки матки, (e) получение кандидатных CGID из классификации на этапе d для получения ранних предикторов рака шейки матки в виде биомаркеров метилирования ДНК, где указанное измерение метилирования ДНК выполняется с использованием методов, включающих в себя чипы Illumina 27K, 450K или 850K, бисульфитное секвенирование всего генома на платформах, включающих в себя секвенаторы HiSeq, MiniSeq, MiSeq или NextSeq, торрент-секвенирование, секвенирование с помощью иммунопреципитации метилированной ДНК (MeDIP), гибридизацию с чипами олигонуклеотидов, пиросеквенирование ДНК, анализы метилирования на основе масс-спектрометрии (Epityper™) или ПЦР.
В еще одном варианте осуществления настоящее изобретение относится к in vitro способу получения ранних предикторов рака шейки матки, включающему в себя следующие этапы: (a) измерение метилирования образца ДНК, выделенного из образца шейки матки, (b) выполнение статистического анализа результатов измерения метилирования ДНК, полученных на этапе a, (c) определение статуса метилирования ДНК множества независимых геномных положений CG, называемых CG-идентификаторами (CGID), путём выполнения анализа прогрессивных изменений метилирования ДНК (APDMA) профилей метилирования ДНК всего генома, полученных на этапе b, (d) классификация CGID на основе частоты их метилирования ДНК, коррелирующей с прогрессированием предраковой стадии рака шейки матки, (e) получение кандидатных CGID из классификации на этапе d для получения ранних предикторов рака шейки матки в виде биомаркеров метилирования ДНК, где указанный статистический анализ результатов измерения метилирования ДНК включает в себя корреляцию Пирсона, анализ соотношений правильного и ложного обнаружения сигналов (ROC) и иерархический кластерный анализ.
В другом варианте осуществления настоящее изобретение относится к in vitro способу получения ранних предикторов рака шейки матки, включающему в себя следующие этапы: (a) измерение метилирования образца ДНК, выделенного из образца шейки матки, (b) выполнение статистического анализа результатов измерения метилирования ДНК, полученных на этапе a, (c) определение статуса метилирования ДНК множества независимых геномных положений CG, называемых CG-идентификаторами (CGID), путём выполнения анализа прогрессивных изменений метилирования ДНК (APDMA) профилей метилирования ДНК всего генома, полученных на этапе b, (d) классификация CGID на основе частоты их метилирования ДНК, коррелирующей с прогрессированием предраковой стадии рака шейки матки, (e) получение кандидатных CGID из классификации на этапе d для получения ранних предикторов рака шейки матки в виде биомаркеров метилирования ДНК, где указанное прогрессирование предраковой стадии рака шейки матки включает в себя поражения при дисплазии шейки матки на стадиях ДШМ, ДШМ2 и ДШМ3.
В альтернативном варианте осуществления настоящее изобретение относится к in vitro способу получения ранних предикторов рака шейки матки, включающему в себя следующие этапы: (a) измерение метилирования образца ДНК, выделенного из образца шейки матки, (b) выполнение статистического анализа результатов измерения метилирования ДНК, полученных на этапе a, (c) определение статуса метилирования ДНК множества независимых геномных положений CG, называемых CG-идентификаторами (CGID), путём выполнения анализа прогрессивных изменений метилирования ДНК (APDMA) профилей метилирования ДНК всего генома, полученных на этапе b, (d) классификация CGID на основе частоты их метилирования ДНК, коррелирующей с прогрессированием предраковой стадии рака шейки матки, (e) получение кандидатных CGID из классификации на этапе d для получения ранних предикторов рака шейки матки в виде биомаркеров метилирования ДНК, где указанные CGID на основе частоты их метилирования ДНК, коррелирующей с прогрессированием предраковой стадии рака шейки матки, выбраны из группы CGID, как указано в SEQ ID NO:1 - SEQ ID NO:79, и их комбинации. В дополнительном варианте осуществления настоящего изобретения 79 сайтов CGID применимы по отдельности или в комбинации, в качестве ранних предикторов рака шейки матки, они обозначены как биомаркеры метилирования ДНК для раннего выявления рака шейки матки.
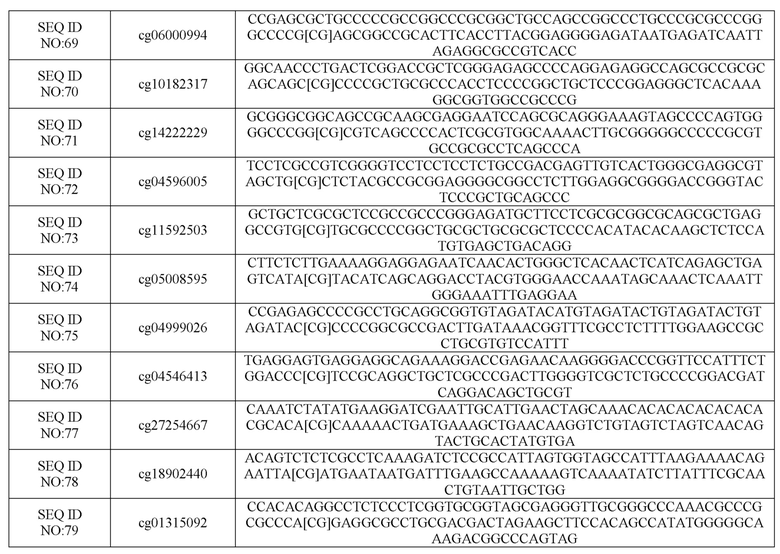
Таблица 1: Выбранные 79 полинуклеотидов, имеющих сайты метилирования CG (CGID), применимые в вариантах осуществления настоящего изобретения.
Последовательности для зонда-идентификатора Illumina для выбранных 79 CGID, используемых в различных вариантах осуществления настоящего изобретения, находятся в Таблице 1, которая включена в настоящую заявку, и включают
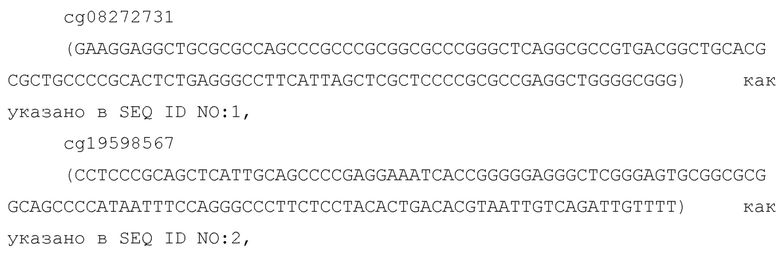









Указанные биомаркеры были включены в окончательный список как прогрессивно метилированные CGID со средним увеличением метилирования на 10% или снижением более чем на 10% во время перехода от стадии ДШМ1 к стадии ДШМ3 и с фоновым метилированием в нормальных клетках (менее 10%) с использованием расчетов, полученных способом APDMA, раскрытым в настоящем изобретении. Метод Illumina использует преимущества последовательностей, фланкирующих локус CG, для генерации уникального идентификатора кластера локуса CG с той же стратегией, что и идентификаторы refSNP NCBI (rs#) в dbSNP.
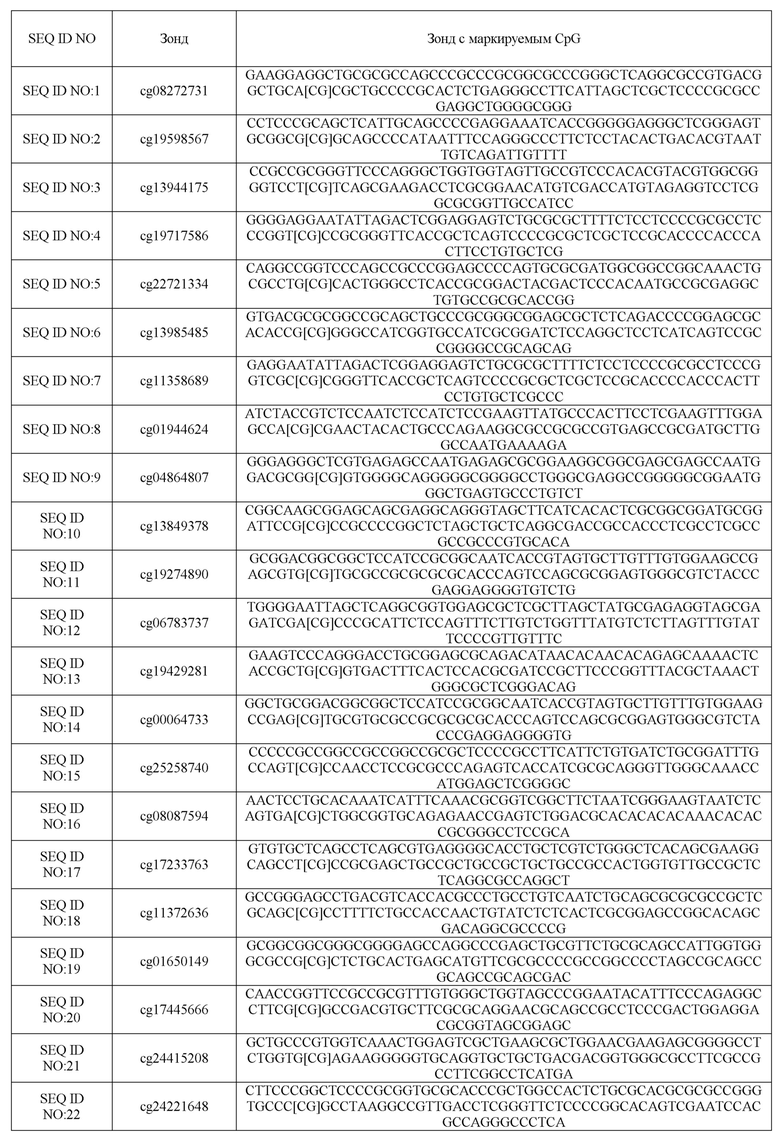
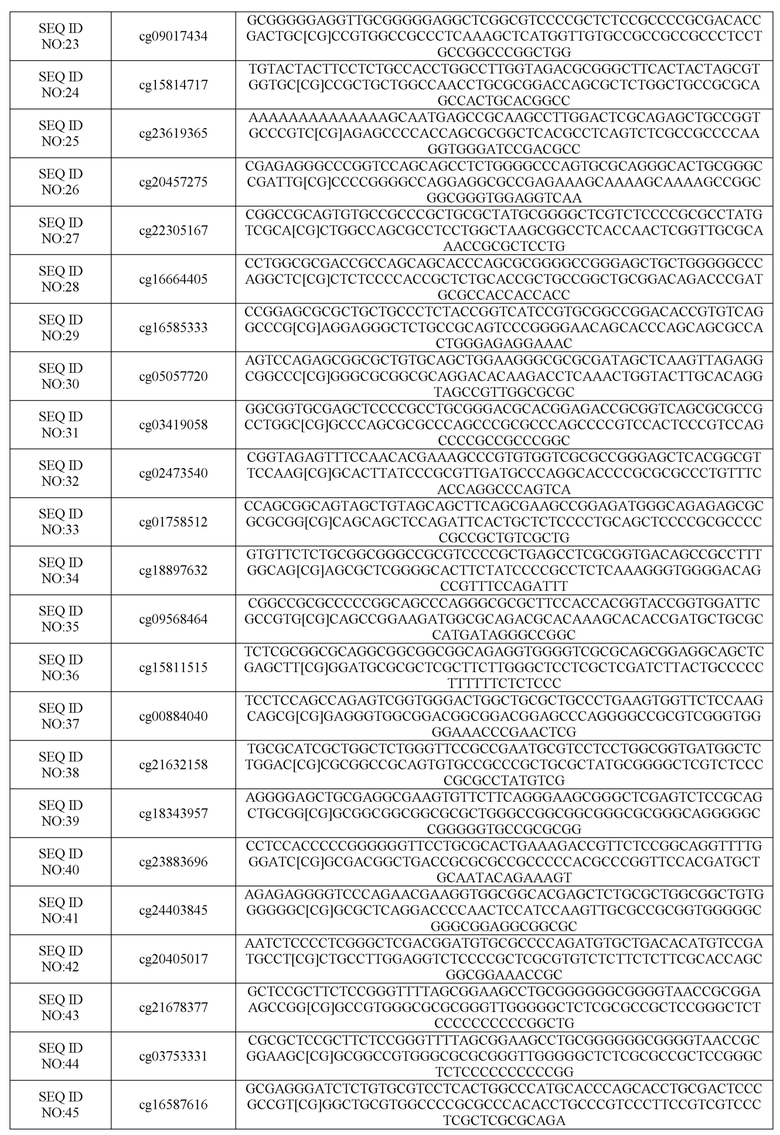
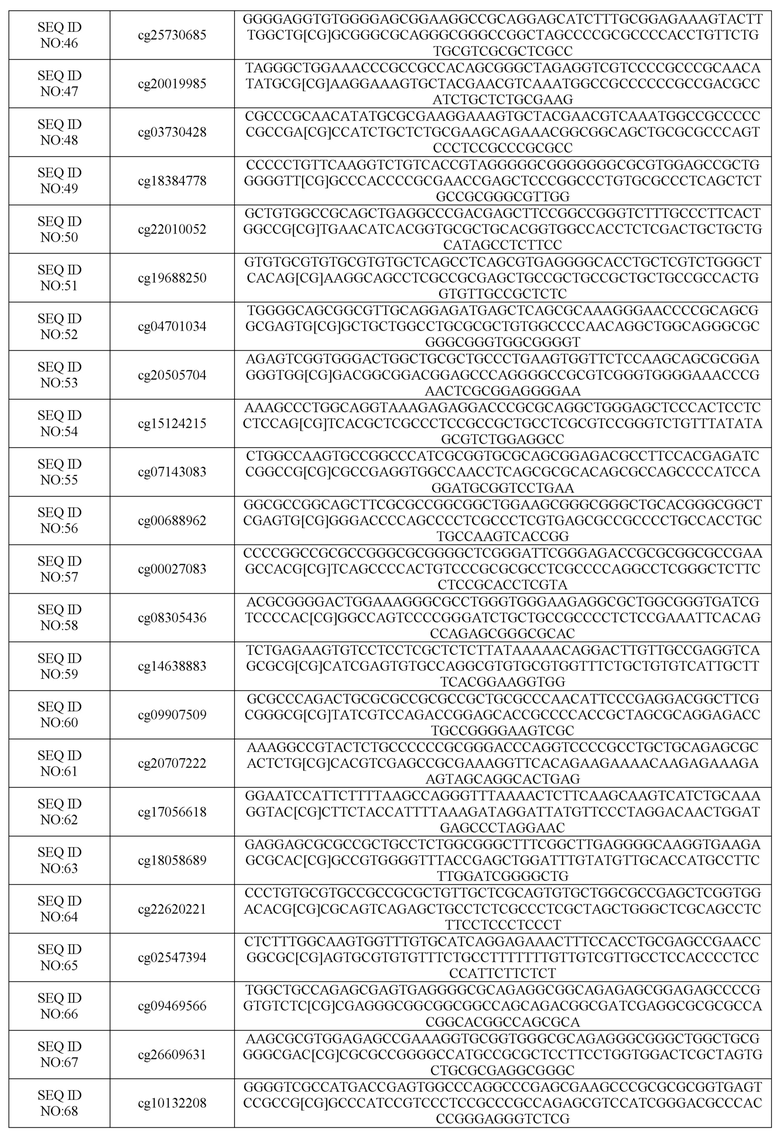
В одном варианте осуществления настоящее изобретение относится к панели биомаркеров метилирования ДНК для скрининга и раннего выявления рака шейки матки, где панель включает в себя CGID, полученные способом APDMA, с последовательностями, выбранными из группы, состоящей из SEQ ID NO:1 - SEQ ID NO:79 и их комбинаций, и, необязательно, указанная панель применяется в комбинации с другими биомаркерами в качестве ранних предикторов рака шейки матки.
В одном варианте осуществления настоящего изобретения биомаркеры метилирования полигенной ДНК представляют собой комбинацию CGID из списка, приведенного ниже в Таблице 2, или короткого подмножества этого списка, такого как пример, приведенный ниже в Таблице 3, для раннего выявления рака шейки матки и риска рак шейки матки у женщин с предраковыми поражениями от ДШМ1 до ДШМ3.
Таким образом, в дополнительном варианте осуществления настоящее изобретение относится к in vitro способу для получения ранних предикторов рака шейки матки, этот способ включает в себя следующие этапы: (a) измерение метилирования образца ДНК, выделенного из образца шейки матки, (b) выполнение статистического анализа результатов измерения метилирования ДНК, полученных на этапе a, (c) определение статуса метилирования ДНК множества независимых геномных положений CG, называемых CG-идентификаторами (CGID), путём выполнения анализа прогрессивных изменений метилирования ДНК (APDMA) профилей метилирования ДНК всего генома, полученных на этапе b, (d) классификация CGID на основе частоты их метилирования ДНК, коррелирующей с прогрессированием предраковой стадии рака шейки матки, (e) получение кандидатных CGID из классификации на этапе d для получения ранних предикторов рака шейки матки в виде биомаркеров метилирования ДНК, где указанные кандидатные CGID в качестве ранних предикторов рака шейки матки в виде биомаркеров метилирования ДНК, где CGID выбраны из группы, как указано в


Таблица 2: Выбранное подмножество полинуклеотидов из Таблицы 1, имеющих сайты метилирования CpG, применимые в вариантах осуществления настоящего изобретения.
Обсуждаемые здесь 16 биомаркеров CGID находятся в Таблице 2, которая включена в данную заявку. Эти включенные в короткий список 16 биомаркеров метилирования ДНК были гиперметилированы на стадиях ДШМ3 и ДШМ1 по сравнению контролем, с наивысшей величиной эффекта (Cohen D> 1,3) между ДШМ3 и контролем и наивысшей корреляцией Спирмена с прогрессированием фаз ДШМ r > 0,4.
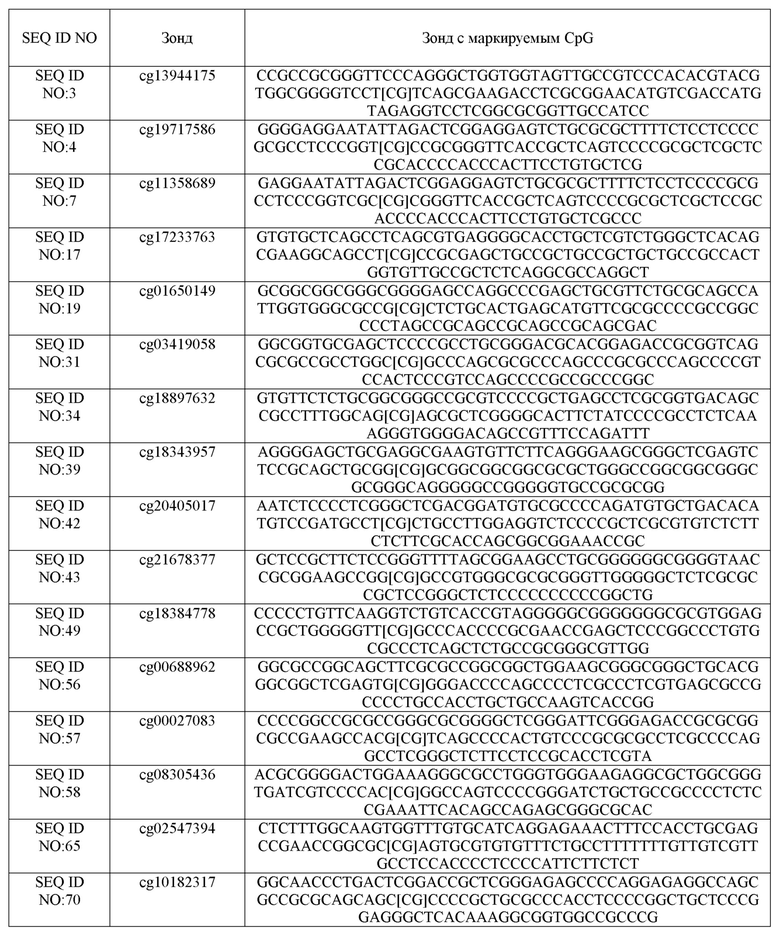
В одном из вариантов осуществления настоящее изобретение относится к комбинации биомаркеров метилирования ДНК для скрининга и раннего выявления рака шейки матки, указанная комбинация включает в себя CGID, полученные с использованием способа APDMA, для выявления рака шейки матки путём измерения уровней метилирования ДНК указанных CGID в ДНК, выделенной из образцов шейки матки, и получение «предиктора метилирования рака шейки матки» с использованием уравнений линейной регрессии и анализа соотношений правильного и ложного обнаружения сигналов (ROC), где CGID выбраны из группы, указанной в SEQ ID NO: 3, SEQ NO: 4, SEQ ID NO: 7, SEQ ID NO: 17, SEQ ID NO: 19, SEQ ID NO: 31, SEQ ID NO: 34, SEQ ID NO: 39, SEQ ID NO: 42, SEQ ID NO: 43, SEQ ID NO: 49, SEQ ID NO: 56, SEQ ID NO: 57, SEQ ID NO: 58, SEQ ID NO: 65, SEQ ID NO: 70 и их комбинации.
Таблица 3: Выбранное подмножество полинуклеотидов из Таблицы 2, имеющих сайты метилирования CpG, используемые в вариантах осуществления изобретения.
Два биомаркера CGID, а именно

как обсуждается здесь, находятся в Таблице 3, которая включена в настоящую заявку. Подмножество в Таблице 3 представляет минимальное количество биомаркеров CGID, которые отличают предраковые поражения ДШМ3 от контроля, идентифицированные с использованием регрессия со штрафом, которая уменьшила количество CGID до 5, с последующей многомерной линейной регрессией с этими 5 CGID в качестве независимых переменных и состоянием ДШМ3 в качестве зависимой переменной. Уравнение линейной регрессии, составленное из взвешенных уровней метилирования этих двух сайтов, было очень значимым для предсказания ДШМ3 (p < 5x10-15).

В одном варианте осуществления настоящее изобретение относится к комбинации биомаркеров метилирования ДНК для скрининга и раннего выявления рака шейки матки, указанная комбинация включает в себя CGID, полученные с использованием способа APDMA, для выявления рака шейки матки путём измерения уровней метилирования ДНК указанных CGID в ДНК, выделенной из образцов шейки матки, и получение «предиктора метилирования рака шейки матки» с использованием уравнений линейной регрессии и анализа соотношений правильного и ложного обнаружения сигналов (ROC), где, где указанные CGID приведены в SEQ ID NO: 3 и SEQ ID NO: 31.
В одном из вариантов осуществления настоящее изобретение относится к набору и способу для выявления рака шейки матки, содержащему средства и реагенты для обнаружения измерений метилирования ДНК панели полигенных биомаркеров метилирования ДНК для рака шейки матки.
В одном из вариантов осуществления настоящее изобретение относится к набору для выявления рака шейки матки, содержащему средства и реагенты для измерения метилирования ДНК биомаркеров CGID из Таблицы 1 и их комбинаций.
В одном из вариантов осуществления настоящее изобретение относится к набору для выявления рака шейки матки, содержащему средства и реагенты для измерения метилирования ДНК CGID и получения предиктора метилирования ДНК рака шейки матки, и инструкцию по применению, где CGID представляют собой SEQ ID NO:1 - SEQ ID NO:79 и их комбинации.
В одном из вариантов осуществления настоящее изобретение относится к набору, состоящему из панели CGID в форме чипа для выявления рака шейки матки, где панель CGID представляют собой последовательности, указанные в SEQ ID NO:1 - SEQ ID NO:79, и их комбинации.
В одном из вариантов осуществления настоящее изобретение относится к набору, в котором используются биомаркеры CGID, раскрытые в настоящем изобретении.
В одном из вариантов осуществления настоящее изобретение относится к набору, использующему анализ метилирования ДНК путём пиросеквенирования для прогнозирования рака шейки матки путём измерения метилирования ДНК CGID, где CGID представляют собой последовательности, указанные в SEQ ID NO:1 - SEQ ID NO:79 и их комбинации.
В одном из вариантов осуществления настоящее изобретение относится к набору, использующему анализ метилирования ДНК путём пиросеквенирования для прогнозирования рака шейки матки с использованием биомаркеров CGID, перечисленных выше, например, с использованием описанных ниже праймеров и стандартных условий реакций пиросеквенирования, рекомендованных производителем (Pyromark, Qiagen):
Для cg03419058:
Прямой (биотинилированный) праймер, указанный в SEQ ID NO:80, с полинуклеотидной последовательностью
GGTTTTTGGGTAGGAAGGATAGTAG.
Обратный праймер, указанный в SEQ ID NO:81, с полинуклеотидной последовательностью
AAACAAATCTAACCCCTAAAAAAAC.
Праймер для пиросеквенирования, указанный в SEQ ID NO:82, с полинуклеотидной последовательностью
CAAACTAAACACACTAAACC.
Для cg13944175:
Прямой праймер, указанный в SEQ ID NO:83, с полинуклеотидной последовательностью
GGGTTTTTAGGGTTGGTGGTA.
Обратный (биотинилированный) праймер, указанный в SEQ ID NO:84, с полинуклеотидной последовательностью
TCCTCATAATAAATAACAACC.
Праймер для пиросеквенирования, указанный в SEQ ID NO:85, с полинуклеотидной последовательностью
TATGTATGTGGTGGGGTT.
В одном из вариантов осуществления настоящее изобретение относится к набору, использующему анализ метилирования ДНК путём пиросеквенирования для прогнозирования рака шейки матки путём измерения метилирования ДНК комбинаций CGID, где прямой биотинилированный праймер имеет последовательность, приведенную в SEQ ID NO:80, обратный праймер имеет последовательность, приведенную в SEQ ID NO:81, и праймер для пиросеквенирования имеет последовательность, приведенную в SEQ ID NO:82.
В одном из вариантов осуществления настоящее изобретение относится к набору, использующему анализ метилирования ДНК путём пиросеквенирования для прогнозирования рака шейки матки путём измерения метилирования ДНК комбинаций CGID, где прямой биотинилированный праймер имеет последовательность, приведенную в SEQ ID NO:83, обратный праймер имеет последовательность, приведенную в SEQ ID NO: 84, и праймер для пиросеквенирования имеет последовательность, приведенную в SEQ ID NO:85.
В одном из вариантов осуществления настоящее изобретение относится к набору, использующему анализ метилирования ДНК с секвенированием полигенных мультиплексных бисульфитных ампликонов для прогнозирования рака шейки матки в ДНК, выделенной из образцов шейки матки, с использованием биомаркеров CGID, перечисленных выше. Например, с использованием описанных ниже праймеров и стандартных условий, которые включают в себя превращение бисульфита, последовательную амплификацию с праймерами, специфичными для мишени (ПЦР1), с последующими штрих-кодирующими праймерами (ПЦР2) и мультиплексное секвенирование в одном секвенаторе Miseq следующего поколения (Illumina), демультиплексирование с использованием программного обеспечения Illumina, извлечение данных и количественную оценку метилирования с использованием стандартных методов анализа метилирования, таких как Methylkit, с последующим расчетом взвешенной оценки метилирования ДНК и прогнозированием рака.
Первую ПЦР проводят следующим образом:
Для CGID cg03419058:
Прямой праймер, указанный в SEQ ID NO:80, с полинуклеотидной последовательностью
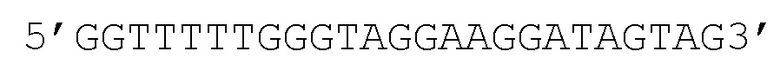
Обратный праймер, указанный в SEQ ID NO:81, с полинуклеотидной последовательностью
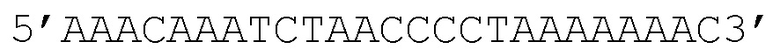
Для CGID cg13944175:
Прямой праймер, указанный в SEQ ID NO:83, с полинуклеотидной последовательностью
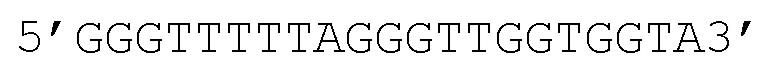
Обратный праймер, указанный в SEQ ID NO:84, с полинуклеотидной последовательностью

Для штрих-кодирования (индексации) образцов в настоящем изобретении использовали вторую реакция ПЦР со следующими праймерами:
Прямой праймер, указанный в SEQ ID NO:86, с полинуклеотидной последовательностью
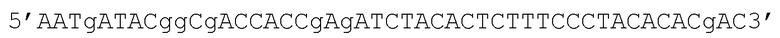
Штрих-кодирующий (обратный) праймер, указанный в SEQ ID NO:87, с полинуклеотидной последовательностью
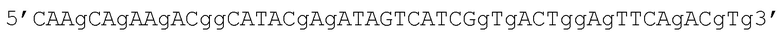
(где красные основания являются индексом; и используется 1200 вариантов этого индекса).
В одном из вариантов осуществления настоящее изобретение относится к набору, использующему анализ метилирования с помощью бисульфитного секвенирования с мультиплексной целевой амплификацией на секвенаторе следующего поколения для выявления рака шейки матки путём измерения уровней метилирования ДНК комбинаций CGID, где CGID указаны в SEQ ID NO:1 - SEQ ID NO:79 и их комбинации.
В другом варианте осуществления настоящее изобретение относится к набору, использующему анализ метилирования с помощью бисульфитного секвенирования с мультиплексной целевой амплификацией на секвенаторе следующего поколения для выявления рака шейки матки путём измерения уровней метилирования ДНК комбинаций CGID, где CGID, как указано в SEQ ID NO:3 содержит праймеры, указанный в SEQ ID NO:88, с полинуклеотидной последовательностью
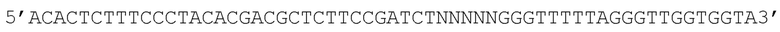
для прямого праймера и указанный в SEQ ID NO:89 с полинуклеотидной последовательностью
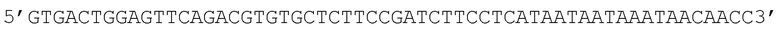
для обратного праймера.
В другом варианте осуществления настоящее изобретение относится к набору, использующему анализ метилирования с помощью бисульфитного секвенирования с мультиплексной целевой амплификацией на секвенаторе следующего поколения для выявления рака шейки матки путём измерения уровней метилирования ДНК комбинаций CGID, где CGID, как указано в SEQ ID NO:31 содержит праймеры, указанный в SEQ ID NO:90 с полинуклеотидной последовательностью
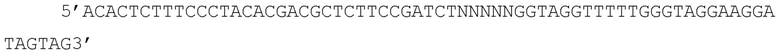
для прямого праймера и указанный в SEQ ID NO:91 с полинуклеотидной последовательностью
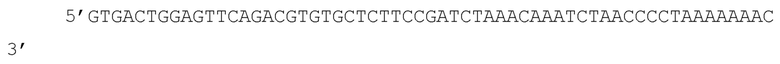
для обратного праймера.
В одном из вариантов осуществления настоящее изобретение относится к применению анализа соотношений правильного и ложного обнаружения сигналов (ROC) для выявления рака путём определения порогового значения между раком шейки матки и нормальной шейкой матки с использованием взвешенных измерений метилирования ДНК биомаркеров CGID, приведенных в Таблице 1, и их комбинаций или подмножества этих идентификаторов CGID, такие например, как приведенные в Таблице 2 и их комбинации. Образцы выше порогового значения классифицируются как раковые.
В одном из вариантов осуществления настоящее изобретение относится к применению иерархического кластерного анализа для прогнозирования рака с использованием в получении положительного результата раннего обнаружения рака с использованием измерений метилирования биомаркеров CGID, перечисленных в Таблице 1, и их комбинаций.
В одном из вариантов осуществления настоящее изобретение относится к набору, использующему анализ метилирования выделенной из образца ДНК на основе масс-спектрометрии (Epityper™) или ПЦР для выявления рака путём измерения уровней метилирования ДНК комбинаций CGID в панели биомаркеров метилирования ДНК для скрининга и раннего выявления рака шейки матки, где панель включает в себя CGID, полученные способом APDMA, имеющие последовательности, выбранные из группы, состоящей из SEQ ID NO:1 - SEQ ID NO:79 и их комбинаций, и, необязательно, указанная панель используется в комбинации с другими биомаркерами в качестве ранних предикторов рака шейки матки.
В одном из вариантов осуществления настоящее изобретение относится к применению уравнения многомерной линейной регрессии или нейросетевого анализа для расчета показателя метилирования, прогнозирующего рак шейки матки, с использованием измерений метилирования ДНК комбинаций CGID в панели биомаркеров метилирования ДНК для скрининга и раннего выявления рака шейки матки, где панель включает в себя CGID, полученные способом APDMA, с последовательностями, выбранными из группы, состоящей из SEQ ID NO:1 - SEQ ID NO:79 и их комбинаций, и, необязательно, указанная панель используется в комбинации с другими биомаркерами в качестве ранних предикторов рака шейки матки.
В одном из вариантов осуществления настоящее изобретение относится к применению уравнения многомерной линейной регрессии или нейросетевого анализа для расчета показателя метилирования, прогнозирующего рак шейки матки, с использованием измерений метилирования ДНК комбинаций CGID в комбинации биомаркеров метилирования ДНК для скрининга и раннего выявления рака шейки матки, указанная комбинация включает в себя CGID, полученные с использованием способа APDMA, для выявления рака шейки матки путём измерения уровней метилирования ДНК указанных CGID в ДНК, выделенной из образцов шейки матки, и получения «предиктора метилирования рака шейки матки» с использованием уравнений линейной регрессии и анализа соотношений правильного и ложного обнаружения сигналов (ROC), где CGID выбраны из группы, состоящей из SEQ ID NO:3, SEQ NO:4, SEQ ID NO:7, SEQ ID NO:17, SEQ ID NO:19, SEQ ID NO:31, SEQ ID NO:34, SEQ ID NO:39, SEQ ID NO:42, SEQ ID NO:43, SEQ ID NO:49, SEQ ID NO:56, SEQ ID NO:57, SEQ ID NO:58, SEQ ID NO:65, SEQ ID NO:70 и их комбинаций.
В альтернативном варианте осуществления настоящее изобретение относится к применению уравнения многомерной линейной регрессии или нейросетевого анализа для расчета показателя метилирования, прогнозирующего рак шейки матки, с использованием измерений метилирования ДНК комбинаций CGID в комбинации биомаркеров метилирования ДНК, где указанные CGID имеют последователности, указанные в SEQ ID NO:3 и SEQ ID NO: 31.
В одном из вариантов осуществления настоящее изобретение относится к применению анализа соотношений правильного и ложного обнаружения сигналов (ROC) для определения порога «показателя метилирования», позволяющего дифференцировать рак шейки матки от нераковой ткани шейки матки с использованием измерений комбинаций метилирования ДНК в панели биомаркеров метилирования ДНК для скрининга и раннего выявления рака шейки матки, где панель включает в себя CGID, полученные способом APDMA, имеющие последовательности, выбранные из группы, состоящей из SEQ ID NO:1 - SEQ ID NO:79 и их комбинаций, и, необязательно, указанная панель используется в комбинация с другими биомаркерами в качестве ранних предикторов рака шейки матки.
В одном из вариантов осуществления настоящее изобретение относится к применению анализа соотношений правильного и ложного обнаружения сигналов (ROC) для определения порогового значения «показателя метилирования», позволяющего дифференцировать рак шейки матки от нераковой ткани шейки матки с использованием измерений комбинаций метилирования ДНК в комбинации биомаркеров метилирования ДНК для скрининга и раннего выявления рака шейки матки, указанная комбинация включает в себя CGID, полученные с использованием способа APDMA, для выявления рака шейки матки путём измерения уровней метилирования ДНК указанных CGID в ДНК, выделенной из образцов шейки матки, и получения «предиктора метилирования рака шейки матки» с использованием уравнения линейной регрессии и анализа соотношений правильного и ложного обнаружения сигналов (ROC), где идентификаторы CGID выбраны из группы, состоящей из SEQ ID NO:3, SEQ NO:4, SEQ ID NO:7, SEQ ID NO:17, SEQ ID NO:19, SEQ ID NO:31, SEQ ID NO:34, SEQ ID NO:39, SEQ ID NO:42, SEQ ID NO:43, SEQ ID NO:49, SEQ ID NO:56, SEQ ID NO:57, SEQ ID NO:58, SEQ ID NO:65, SEQ ID NO:70 и их комбинаций.
В альтернативном варианте осуществления настоящее изобретение относится к применению анализа соотношений правильного и ложного обнаружения сигналов (ROC) для определения порогового значения «показателя метилирования», позволяющего дифференцировать рак шейки матки от нераковой ткани шейки матки с использованием измерений комбинаций метилирования ДНК в комбинации биомаркеров метилирования ДНК, где указанные CGID имеют последовательности, указанные в SEQ ID NO:3 и SEQ ID NO:31.
В одном из вариантов осуществления настоящее изобретение относится к реализованному на компьютере способу получения кандидатных биомаркеров метилирования ДНК для раннего выявления для диагностики рака шейки матки, данный способ включает в себя: предоставление данных о метилировании ДНК по всему геному для множества независимых геномных положений CG, CGID генома человека; обработку данных о метилировании ДНК по всему геному путём нормализации и получения нормализованных бета-значений метилирования ДНК; вычисление корреляции Спирмена с нормализованными бета-значениями метилирования ДНК между стадиями прогрессирования предраковых состояний и неизмененными клетками шейки матки; получение кандидатных CGID при помощи анализа прогрессирующих изменений метилирования ДНК (APDMA) для получения кандидатных биомаркеров метилирования ДНК для ранней диагностики рака шейки матки.
Примеры
Следующие ниже примеры приводятся для иллюстрации настоящего изобретения, и не должны рассматриваться как ограничивающие объем настоящего изобретения.
Пример 1: Способ анализа прогрессирующих изменений метилирования ДНК (APDMA) для идентификации и получения положений CG (CGID), уровень метилирования которых является ранним предиктором рака шейки матки.
Настоящее изобретение направлено на решение одной из важнейших задач скрининга рака шейки матки, заключающейся в нахождении надежных биомаркеров, которые обеспечивают высокоточную и чувствительную оценку риска, которая может служить ориентиром для раннего вмешательства и лечения. Обычные подходы заключаются в использовании логистической регрессии случай-контроль данных метилирования ДНК в масштабе всего генома для выявления участков, которые либо более, либо менее метилированы в раковых клетках по сравнению с контрольными. Однако хорошо известно, что многие статистически значимые изменения метилирования ДНК при раке, обнаруженные этими методами, являются гетерогенными, и многие из них развиваются на поздних стадиях прогрессирования рака и, следовательно, имеют очень ограниченную ценность для раннего выявления, поскольку они уменьшаются, когда частота раковых клеток в образце является низкой. Более того, количественные различия в профилях метилирования, а не однозначные различия могут быть стерты в смеси нормальных и раковых клеток. Как хорошо известно, метилирование ДНК является бинарным свойством, что означает, что данная клетка либо метилирована, либо у нее в определенном положении в геноме не находится CG.
В этом примере настоящее изобретение относится к выбранным метилированным CGID как фундаментальной характеристике рака шейки матки, которые почти равномерно метилированы в образцах рака шейки матки, но никогда не метилируются в нормальной ткани, и, несмотря на то, что они однозначны для рака шейки матки, они возникают очень рано на предраковых стадиях в среде нормальных клеток, и их частота постепенно увеличивается от стадий ДШМ1 к ДШМ3, прогрессирующих к раку шейки матки. Было обнаружено, что метилированные CGID, которые однозначно различаются между нормальными и раковыми тканями, обнаруживаются даже тогда, когда раковые клетки обнаруживаются с низкой частотой в образце, путём глубокого секвенирования ДНК, преобразованной бисульфитом, которое обеспечивает разрешение одной молекулы ДНК. Частота молекул с метилированным CGID представляет собой долю раковых клеток в образце. Измерения метилирования таких CGID другими методами также могут определять частоту раковых клеток в образце и могут использоваться в качестве биомаркеров метилирования ДНК для оценки риска и прогнозирования рака шейки матки в образце.
Клинически известно, что часть предраковых поражений ДШМ перерастает в рак шейки матки, поэтому они являются особенно уникальным моментом для обнаружения ранних изменений метилирования ДНК при раке. Раннее прогнозирование того, у кого разовьется рак шейки матки, имеет огромное клиническое значение. Настоящее изобретение относится к способу получения таких биомаркеров метилирования ДНК для раннего выявления, характеризующихся следующими техническими признаками: во-первых, метилированные CGID, которые были однозначно характерны для ранних раковых клеток, были однородно неметилированы в нормальной ткани шейки матки; во-вторых, эти CGID нечасто метилировались в ранних предраковых образцах; в-третьих, частота этих первичных метилированных CGID должна увеличиваться с прогрессированием предраковых стадий от ДШМ1 до ДШМ3, так как это связано с повышенным риском рака шейки матки у женщин с поражениями ДШМ3; и в-четвертых, поскольку метилирование этих CGID является основной характеристикой рака шейки матки, эти CGID должны быть равномерно многочисленными в образцах рака шейки матки. В этом примере было обнаружено, что определенные CGID, метилирование которых увеличивается с прогрессированием стадий ДШМ от ДШМ1 к ДШМ3, повсеместно метилированы в образцах рака шейки матки, в то время как было обнаружено, что они однородно неметилированы в нормальной ткани, как описано здесь. Таким образом, раскрытый здесь способ настоящего изобретения относится к панели кандидатных биомаркеров CGID для раннего выявления рака шейки матки у женщин, особенно с предраковыми поражениями.
Следующие этапы способа прогрессивных изменений метилирования ДНК (APDMA) были выполнены для определения биомаркеров CGID, состояние метилирования которых выявляет ранний рак шейки матки, как показано на Фигуре 1.
Образцы шейки матки
В настоящем изобретении использовались образцы шейки матки, взятые у женщин, направленных на кольпоскопическое обследование в больницу Университета Мак-Гилла из-за аномального результата скрининга на рак шейки матки или для начального лечения поражения шейки матки (19). В кратком изложении, в период с июня 2015 г. по апрель 2016 г. были обследованы 643 женщины в возрасте 16-70 лет. Образцы были протестированы на наличие ДНК ВПЧ канцерогенных типов с помощью теста Roche cobas® 4800 HPV Test, который выявляет ВПЧ1 и ВПЧ18 по отдельности, а также 12 других типов, связанных с высоким риском рака шейки матки (ВПЧ 31, 33, 35, 39, 45, 51, 52, 56, 58, 59, 66 и 68) с получением объединенного результата. Результаты цитологического исследования классифицировали в соответствии с классификацией Bethesda как NILM: внутриэпителиальные поражения и злокачественные образования отсутствуют; ASC-US: атипичные клетки плоского эпителия неясного значения; ASC-H атипичные клетки плоского эпителия, нельзя исключить высокую степень плоскоклеточного интраэпителиального поражения (HSIL); LSIL: низкая степень плоскоклеточного интраэпителиального поражения; HSIL: высокая степень плоскоклеточного интраэпителиального поражения; AGC: атипичные клетки железистого эпителия; и рак (20). Делали биопсию патологических тканей шейки матки, и гистологические результаты старшие патологоанатомы Университета Мак-Гилла оценивали как нормальные, ДШМ1, ДШМ2, ДШМ3 или инвазивный рак. Исследование получило этическое одобрение институциональных наблюдательных советов Университета Макгилла и Еврейской больницы общего профиля. Участники исследования предоставили письменное информированное согласие.
Набор образцов включал в себя 186 случайно выбранных образцов женщин, собранных врачом. Из этих образцов 50 были ДШМ1, 40 ДШМ2 и 42 ДШМ3 и 54 образцов с нормальным результатом биопсии.
Выделение ДНК и анализ метилирования генома
ДНК выделяли из исходных образцов расслоенных цервикальных клеток, суспендированных в жидком цитологическом растворе PreservCyt (PreservCyt, Hologic Inc., Миссиссауга).
ДНК, выделенную с использованием набора для экстракции ДНК Qiagen, подвергали обработке бисульфитом и гибридизации с чипами Illumina Epic с использованием стандартных процедур, описанных производителем в инновационном центре Genome Quebec в Монреале. Чипы Epic обеспечивают превосходное покрытие репертуара промоторов и энхансеров человека, охватывая все известные области, регулирующие транскрипцию (21).
Нормализация и получение нормализованных показателей метилирования ДНК (бета) для всех образцов
Образцы рандомизировали по препарату и положению на чипах, и все образцы гибридизовали и сканировали одновременно для смягчения групповых эффектов, как рекомендовано инновационным центром McGill Genome в Квебеке в соответствии с руководством пользователя технологии Illumina Infinium HD. Гибридизация и сканирование чипов Illumina были выполнены инновационном центре McGill Genome в Квебеке в соответствии с инструкциями производителя. Чипы Illumina проанализировали с использованием пакета ChAMP Bioconductor в версии R Morris et al., 2014 (25). Файлы IDAT использовали в качестве входных данных в функции champ.load с использованием параметров контроля качества и нормализации minfi. Необработанные данные были отфильтрованы для зондов со значением обнаружения P > 0,01 как минимум в одном образце. Настоящий метод отфильтровывает зонды на X- или Y-хромосоме для смягчения половых эффектов и зонды с ОНП, как указано в Marzouka et al., 2015 (24), а также зонды, которые выравниваются по нескольким местоположениям, как указано в Marzouka et al., 2015 (24). Групповые эффекты анализировали на ненормализованных данных с помощью функции champ.svd. Пять из первых 6 основных компонентов были связаны с группой и серией (препараты). Нормализация внутри чипа для корректировки данных на предмет погрешности, вносимой конструкцией зонда Infinium типа 2, была выполнена с использованием квантильной нормализации бета-смеси (BMIQ) с функцией champ.norm (norm = «BMIQ») (25). Затем групповые эффекты корректировали после нормализации BMIQ с использованием функции champ.runcombat.
Выявление CGID, частота метилирования которых коррелирует с прогрессированием ДШМ
Затем в настоящем способе использовали бета-значения нормализованных данных, скорректированных по группам, для вычисления корреляции Спирмена между стадиями ДШМ (с кодами стадий 0 для нетрансформированных здоровых контрольных клеток шейки матки и 1-3 для стадий ДШМ от ДШМ1 до ДШМ3) с использованием скорректированной функции Спирмена в версии R и корректировки для множественных испытаний с использованием метода «fdr» Бенджамини Хохберга (скорректированное значение P (Q) < 0,05). Уровни метилирования 7715 CGID достоверно коррелировали (q > 0,05) с прогрессированием предраковых стадий ДШМ от 1 до 3 (см. Фигуру 2). Большинство сайтов были гиперметилированы, когда предраковые поражения прогрессировали от нормального до стадии ДШМ1 и ДШМ3, в то время как небольшая часть была гипометилирована (см. фиг. 2).
Составление короткого списка кандидатных CGID
Чтобы определить положения CGID, которые соответствуют предположениям способа APDMA, 79 прогрессивно метилированных CGID были включены в короткий список со средним увеличением метилирования на 10% или снижением более чем на 10% во время перехода от ДШМ1 к ДШМ3 и фоновым метилированием в нормальных клетках (менее 10%) (см. Таблицу 1 выше). При помощи настоящего способа затем проверяли, одинаково ли эти CGID идентифицируют рак шейки матки в общедоступных данных о метилировании ДНК по всему геному Illumina 450K из 270 образцов рака шейки матки (см. GSE68339). На основе протестированного метилирования ДНК CGID, при помощи настоящего способа затем сгенерировали карту интенсивности с этими 79 CGID, частота метилирования которых увеличивалась во время прогрессирования предраковых стадий шейки матки, которые были получены с помощью описанного здесь способа APDMA. Указанная карта интенсивности показала, что эти 79 CGID демонстрируют совершенно разные профили метилирования ДНК при раке шейки матки и в нормальной шейке матки. Явное большинство сайтов были полностью неметилированы в нормальной ткани и сильно метилированы в раковой ткани, в то время как небольшое количество сайтов было метилировано в нормальной ткани и неметилировано при раке шейки матки (см. Фигуру 3). Таким образом, настоящий способ относится к этим гиперметилированным CGID в качестве предпочтительных биомаркеров, поскольку даже низкая частота метилирования четко выявляется на фоне полностью неметилированных молекул.
Пример 2: Выявление набора биомаркеров полигенного метилирования ДНК для раннего выявления рака шейки матки.
В настоящем описании дополнительно включены 16 CGID из списка, полученного и раскрытого в первом примере и в Таблице 1, где указанные 16 CGID были гиперметилированы при стадиях ДШМ3 и ДШМ1 по сравнению с контролем, имели наибольшую величину эффекта (Cohen D > 1,3) между ДШМ3 и контролем и наивысшую корреляцию Спирмена с прогрессированием фаз ДШМ r>0,4. (см. Таблицу 2 выше).
Затем, чтобы получить минимальное количество CGID, необходимое для дифференциации предраковых поражений ДШМ3 от контроля, в настоящем способе была применена регрессия со штрафом, которая уменьшила количество CGID до 5. В соответствии с настоящим способом затем проводили многомерную линейную регрессию с этими 5 CGID в качестве независимых переменных и стадией ДШМ3 в качестве зависимой переменной. Два CGID оставались значимыми (см. Таблицу 3 выше). Уравнение линейной регрессии, составленное из взвешенных уровней метилирования этих двух сайтов, было очень значимым для прогнозирования ДШМ3 (p <5x10-15).
Пример 3: Применение бигенных маркеров метилирования ДНК для выявления рака шейки матки.
Затем в настоящем изобретении впервые был валидирован бигенный маркер метилирования ДНК (cg03419058; cg13944175) в общедоступной базе данных метилирования ДНК 450K рака шейки матки (см. GSE68339). Модель двухвариантной линейной регрессии с раком шейки матки в качестве зависимой переменной и уровнем метилирования двух CGID (cg03419058; cg13944175) в качестве независимых переменных оказалась очень значимой (p <2,2x10-16, F = 8703, R = 0,9873). ROC для показателей метилирования (рассчитанных с использованием уравнения линейной регрессии, как показано на Фигуре 4A) сравнивали путём вычисления их площади под кривой (AUC) (см. Фигуре 4B). Чувствительность и специфичность показателя бигенного метилирования для различения рака шейки матки и нормальной ткани шейки матки составила 1 (см. Фигуру 4C).
Таким образом, описанные выше биомаркеры метилирования ДНК и рассчитанный показатель метилирования применимы для скрининга и раннего выявления рака шейки матки у женщин из группы риска, а также в целом здоровой популяции женщин с использованием образцов шейки матки, взятых при рутинном гинекологическом осмотре из мазков по Папаниколау.
Пример 4: Применение бигенных биомаркеров метилирования ДНК для измерения показателей метилирования рака шейки матки в отдельных образцах, полученных от здоровых пациентов (контроль), пациентов со стадиями ДШМ от ДШМ1 до ДШМ3 и пациентов с раком шейки матки.
Показатель метилирования (прогноз рака шейки матки) рассчитывали с использованием уравнения, представленного на Фигуре 4A, для каждого из отдельных образцов из контроля, стадий от ДШМ1 до ДШМ3 (из когорты Мак-Гилла, описанной выше в Примере 1) и рака шейки матки (см. GSE68339) (см. Фигуру 5A), (средние значения для различных групп см. на Фигуре 5B). Результаты демонстрируют увеличение показателей метилирования при распространенных предраковых поражениях, как ожидалось на основании клинических наблюдений повышенного риска рака шейки матки с прогрессированием стадий ДШМ. Показатели метилирования могут использоваться для скрининга женщин с поражениями ДШМ на предмет риска развития рака шейки матки.
Пример 5: Корреляция Спирмена показателя метилирования и прогрессирования предракового состояния шейки матки в рак шейки матки.
Проводили корреляционный анализ Спирмена между показателями метилирования образцов шейки матки, полученных от здоровых пациентов, пациентов с предраковыми стадиями от ДШМ1 до ДШМ3 и пациентов с раком шейки матки (контроль, n = 54; ДШМ1, n = 50; ДШМ2, n = 40; ДШМ3, n = 42; рак шейки матки, n = 270). Результаты демонстрируют очень значимую корреляцию (p<2,2x10-16 и r = 0,88) между показателем метилирования бигенного маркера и прогрессированием от предракового состояния к злокачественному состоянию (см. Фигуру 6).
Пример 6: Валидация биомаркера метилирования (cg13944175) для выявления рака шейки матки.
Поскольку данные только для одного биомаркера CGID были доступны в базе данных о раке шейки матки TCGA, в настоящем изобретении рассчитывали показатель метилирования для рака шейки матки с использованием уравнения линейной регрессии с данными метилирования ДНК только для указанного CGID, cg13944175. Корреляция Спирмена была рассчитана между стадией прогрессирования рака и показателем метилирования (см. статистику на Фигуре 7A и график корреляции на Фигуре 7B). В настоящем изобретении образцы ДШМ1-ДШМ3 были получены из когорты Мак-Гилла, как уже было описано в настоящей заявке в Примере 1, и значение показателя основано на присвоенной шкале: Контроль: 0, ДШМ1-3: 1-3, соответственно, и рак шейки матки: 4.
Пример 7: Применение бигенного биомаркера метилирования для выявления рака шейки матки в предраковых образцах шейки матки.
Бигенный биомаркер метилирования использовали для прогнозирования того, в каких образцах со стадиями ДШМ от ДШМ1 до ДШМ3 ДШМ будет прогрессировать до рака шейки матки. Показатели метилирования рассчитывали для каждого образца на основе значений метилирования для двух сайтов CG, полученных из данных чипов Epic. Используя пороговое значение для рака, вычисленное из сравнения образцов рака шейки матки и здоровых образцов шейки матки (см. Фигуру 3), делали прогноз для каждого из образцов (см. Фигуру 8A). Доля образцов, которые, по прогнозам, станут злокачественными, как и ожидалось, увеличивалась с нескольких образцов в образцах ДШМ1 до 60% в образцах ДШМ3, (см. Фигуру 8B).
Хотя настоящее изобретение было объяснено на примере его предпочтительного варианта осуществления, следует понимать, что можно сделать множество других возможных модификаций и изменений, не выходящих за рамки сущности и объема настоящего изобретения.
Преимущества
Эти новые биомаркеры метилирования ДНК могут быть разработаны в качестве диагностического набора для ранней и точной диагностики рака шейки матки человека. Они являются прямыми индикаторами клеточных изменений во время инициации и развития рака шейки матки и представляют фундаментальную характеристику рака шейки матки, являясь почти однородно метилироваными в образцах рака шейки матки, но никогда не метилируясь в нормальной ткани, и их частота метилирования прогрессивно увеличивается от стадий ДШМ1 до ДШМ3 и до злокачественной стадии. Эти биомаркеры дополняют патологоанатомическую оценку для точного раннего выявления рака шейки матки при поражениях ДШМ, а также служат в качестве биомаркера для раннего выявления и прогнозирования риска развития рака шейки матки у бессимптомных женщин. Эти биомаркеры являются полезным дополнением для уже существующих эпигенетических маркеров метилирования ДНК, которые играют важную роль в регуляции генов, и могут использоваться в форме CGID в качестве инструмента диагностики. Эти биомаркеры могут обеспечить быстрый, дешевый, точный, надежный и высокопроизводительный диагностический набор для точной, ранней и пока еще не осуществимой диагностики рака шейки матки человека на еще недоступных предраковых стадиях.
Ссылки
1. El-Zein M, Richardson L, Franco EL. Cervical cancer screening of HPV vaccinated populations: Cytology, molecular testing, both or none. J. Clin. Virol. 2015;76:S62 - S68. doi:10.1016/j.jcv.2015.11.020.
2. Boers A, Wang R, van Leeuwen RW, et al. Discovery of new methylation markers to improve screening for cervical intraepithelial neoplasia grade 2/3. Clin. Epigenetics 2016;8(29). doi:10.1186/s13148-016-0196-3.
3. van Baars R, van der Marel J, Snijders PJF, et al. CADM1 and MAL methylation status in cervical scrapes is representative of the most severe underlying lesion in women with multiple cervical biopsies. Int. J. Cancer 2016;138:463 - 471. doi:10.1002/ijc.29706.
4. Verhoef VMJ, Van Kemenade FJ, Rozendaal L, et al. Follow-up of high-risk HPV positive women by combined cytology and bi-marker CADM1/MAL methylation analysis on cervical scrapes. Gynecol. Oncol. 2015;137(1):55 - 59. doi:10.1016/j.ygyno.2015.01.550.
5. Louvanto K, Franco EL, Ramanakumar A V, et al. Methylation of viral and host genes and severity of cervical lesions associated with human papillomavirus type 16. Int. J. Cancer 2014;00(6):1 - 8. doi:10.1002/ijc.29196.
6. Simanaviciene V, Popendikyte V, Gudleviciene Z, Zvirbliene A. Different DNA methylation pattern of HPV16, HPV18 and HPV51 genomes in asymptomatic HPV infection as compared to cervical neoplasia. Virology 2015;484:227 - 233. doi:10.1016/j.virol.2015.06.008.
7. De Strooper LMA, Meijer CJLM, Berkhof J, et al. Methylation analysis of the FAM19A4 gene in cervical scrapes is highly efficient in detecting cervical carcinomas and advanced CIN2/3 lesions. Cancer Prev. Res. 2014;7(6):1251 - 1257. doi:10.1158/1940-6207.CAPR-14-0237.
8. Mersakova S, Nachajova M, Szepe P, Kasajova PS, Halasova E. DNA methylation and detection of cervical cancer and precancerous lesions using molecular methods. Tumor Biol. 2015. doi:10.1007/s13277-015-4197-1.
9. Nikolaidis C, Nena E, Panagopoulou M, et al. PAX1 methylation as an auxiliary biomarker for cervical cancer screening: a meta-analysis. Cancer Epidemiol. 2015;39(5):682 - 686. doi:10.1016/j.canep.2015.07.008.
10. De Strooper LMA, Hesselink AT, Berkhof J, et al. Combined CADM1/MAL methylation and cytology testing for colposcopy triage of high-risk HPV-positive women. Cancer Epidemiol. Biomarkers Prev. 2014;23(9):1933 - 1937. doi:10.1158/1055-9965.EPI-14-0347.
11. De Strooper LMA, Verhoef VMJ, Berkhof J, et al. Validation of the FAM19A4/mir124-2 DNA methylation test for both lavage- and brush-based self-samples to detect cervical (pre)cancer in HPV-positive women. Gynecol. Oncol. 2016. doi:10.1016/j.ygyno.2016.02.012.
12. Louvanto K, Franco EL, Ramanakumar A V, et al. Methylation of viral and host genes and severity of cervical lesions associated with human papillomavirus type 16. Int. J. cancer 2015;136(6):E638-45. doi:10.1002/ijc.29196.
13. De Strooper LM a, van Zummeren M, Steenbergen RDM, et al. CADM1, MAL and miR124-2 methylation analysis in cervical scrapes to detect cervical and endometrial cancer. J. Clin. Pathol. 2014;67:1067 - 1071. doi:10.1136/jclinpath-2014-202616.
14. Feng C, Dong J, Chang W, Cui M, Xu T. The Progress of Methylation Regulation in Gene Expression of Cervical Cancer. Int. J. Genomics 2018;2018.
15. Del Mistro A, Frayle H, Rizzi M, et al. Methylation analysis and HPV genotyping of self-collected cervical samples from women not responding to screening invitation and review of the literature. PLoS One 2017;12(3):1 - 13. doi:10.1371/journal.pone.0172226.
16. Eijsink JJH, Lendvai A, Deregowski V, et al. A four-gene methylation marker panel as triage test in high-risk human papillomavirus positive patients. Int. J. Cancer 2012;130(8):1861 - 1869. doi:10.1002/ijc.26326.
17. Verlaat W, Snoek BC, Heideman DAM, et al. Identification and validation of a 3-gene methylation classifier for HPV-based cervical screening on self-samples. Clin. Cancer Res. 2018:clincanres.3615.2017. doi:10.1158/1078-0432.CCR-17-3615.
18. Cuzick J, Bergeron C, von Knebel Doeberitz M, et al. New technologies and procedures for cervical cancer screening. Vaccine 2012;30(SUPPL.5):F107 - F116. doi:10.1016/j.vaccine.2012.05.088.
19. El-Zein M, Bouten S, Louvanto K, et al. Validation of a new HPV self-sampling device for cervical cancer screening: The Cervical and Self-Sample In Screening (CASSIS) study. Gynecol. Oncol. 2018. doi:https://doi.org/10.1016/j.ygyno.2018.04.004.
20. Smith JHF. Bethesda 2001. Cytopathology 2002;13(1):4 - 10.
21. Moran S, Arribas C, Esteller M. Validation of a DNA methylation microarray for 850,000 CpG sites of the human genome enriched in enhancer sequences. Epigenomics 2016;8(3):389 - 399. doi:10.2217/epi.15.114.
22. Morris TJ, Butcher LM, Feber A, et al. ChAMP: 450k Chip Analysis Methylation Pipeline. Bioinformatics 2014;30(3):428 - 430. doi:10.1093/bioinformatics/btt684.
23. Luttmer R, De Strooper LMA, Berkhof J, et al. Comparing the performance of FAM19A4 methylation analysis, cytology and HPV16/18 genotyping for the detection of cervical (pre)cancer in high-risk HPV-positive women of a gynecologic outpatient population (COMETH study). Int. J. Cancer 2015;138(May 2015):992 - 1002. doi:10.1002/ijc.29824.
24. Wentzensen N, Schiffman M, Palmer T, Arbyn M. Triage of HPV positive women in cervical cancer screening. J. Clin. Virol. 2016;76:S49 - S55. doi:10.1016/j.jcv.2015.11.015.
25. Marzouka, N. A., Nordlund, J., Backlin, C. L., Lonnerholm, G., Syvanen, A. C., & Carlsson Almlof, J. (2015). CopyNumber450kCancer: baseline correction for accurate copy number calling from the 450k methylation array. Bioinformatics. doi:10.1093/bioinformatics/btv652.
26. Morris, T. J., Butcher, L. M., Feber, A., Teschendorff, A. E., Chakravarthy, A. R., WojdaczT.K., & Beck, S. (2014). ChAMP: 450k Chip Analysis Methylation Pipeline. Bioinformatics, 30(3), 428-430. doi:10.1093/bioinformatics/btt684.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> ЭйчКейДжи ЭПИТЕРАПЬЮТИКС ЛИМИТЕД
<120> Биомаркеры метилирования ДНК для раннего выявления рака шейки
матки
<130> TPC53811
<150> US 62/774,994
<151> 2018-12-04
<160> 91
<170> Патентная версия 3.5
<210> 1
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 1
gaaggaggct gcgcgccagc ccgcccgcgg cgcccgggct caggcgccgt gacggctgca
60
cgcgctgccc cgcactctga gggccttcat tagctcgctc cccgcgccga ggctggggcg
120
gg
122
<210> 2
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 2
cctcccgcag ctcattgcag ccccgaggaa atcaccgggg gagggctcgg gagtgcggcg
60
cggcagcccc ataatttcca gggcccttct cctacactga cacgtaattg tcagattgtt
120
tt
122
<210> 3
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 3
ccgccgcggg ttcccagggc tggtggtagt tgccgtccca cacgtacgtg gcggggtcct
60
cgtcagcgaa gacctcgcgg aacatgtcga ccatgtagag gtcctcggcg cggttgccat
120
cc
122
<210> 4
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 4
ggggaggaat attagactcg gaggagtctg cgcgcttttc tcctccccgc gcctcccggt
60
cgccgcgggt tcaccgctca gtccccgcgc tcgctccgca ccccacccac ttcctgtgct
120
cg
122
<210> 5
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 5
caggccggtc ccagccgccc ggagccccag tgcgcgatgg cggccggcaa actgcgcctg
60
cgcactgggc ctcaccgcgg actacgactc ccacaatgcc gcgaggctgt gccgcgcacc
120
gg
122
<210> 6
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 6
gtgacgcgcg gccgcagctg cccgcgggcg gagcgctctc agaccccgga gcgcacaccg
60
cggggccatc ggtgccatcg cggatctcca ggctcctcat cagtccgccg gggccgcagc
120
ag
122
<210> 7
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 7
gaggaatatt agactcggag gagtctgcgc gcttttctcc tccccgcgcc tcccggtcgc
60
cgcgggttca ccgctcagtc cccgcgctcg ctccgcaccc cacccacttc ctgtgctcgc
120
cc
122
<210> 8
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 8
atctaccgtc tccaatctcc atctccgaag ttatgcccac ttcctcgaag tttggagcca
60
cgcgaactac actgcccaga aggcgccgcg ccgtgagccg cgatgcttgg ccaatgaaaa
120
ga
122
<210> 9
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 9
gggagggctc gtgagagcca atgagagcgc ggaaggcggc gagcgagcca atggacgcgg
60
cggtggggca gggggcgggg cctgggcgag gccgggggcg gaatgggctg agtgccctgt
120
ct
122
<210> 10
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 10
cggcaagcgg agcagcgagg cagggtagct tcatcacact cgcggcggat gcggattccg
60
cgccgccccg gctctagctg ctcaggcgac cgccaccctc gcctcgccgc cgcccgtgca
120
ca
122
<210> 11
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 11
gcggacggcg gctccatccg cggcaatcac cgtagtgctt gtttgtggaa gccgagcgtg
60
cgtgcgccgc gcgcgcaccc agtccagcgc ggagtgggcg tctacccgag gaggggtgtc
120
tg
122
<210> 12
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 12
tggggaatta gctcaggcgg tggagcgctc gcttagctat gcgagaggta gcgagatcga
60
cgcccgcatt ctccagtttc ttgtctggtt tatgtctctt agtttgtatt ccccgttgtt
120
tc
122
<210> 13
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 13
gaagtcccag ggacctgcgg agcgcagaca taacacaaca cagagcaaaa ctcaccgctg
60
cggtgacttt cactccacgc gatccgcttc ccggtttacg ctaaactggg cgctcgggac
120
ag
122
<210> 14
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 14
ggctgcggac ggcggctcca tccgcggcaa tcaccgtagt gcttgtttgt ggaagccgag
60
cgtgcgtgcg ccgcgcgcgc acccagtcca gcgcggagtg ggcgtctacc cgaggagggg
120
tg
122
<210> 15
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 15
cccccgccgg ccgccggccg cgctccccgc cttcattctg tgatctgcgg atttgccagt
60
cgccaacctc cgcgcccaga gtcaccatcg cgcagggttg ggcaaaccat ggagctcggg
120
gc
122
<210> 16
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 16
aactcctgca caaatcattt caaacgcggt cggcttctaa tcgggaagta atctcagtga
60
cgctggcggt gcagagaacc gagtctggac gcacacacac aaacacaccg cgggcctccg
120
ca
122
<210> 17
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 17
gtgtgctcag cctcagcgtg aggggcacct gctcgtctgg gctcacagcg aaggcagcct
60
cgccgcgagc tgccgctgcc gctgctgccg ccactggtgt tgccgctctc aggcgccagg
120
ct
122
<210> 18
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 18
gccgggagcc tgacgtcacc acgccctgcc tgtcaatctg cagcgcgcgc cgctcgcagc
60
cgccttttct gccaccaact gtatctctca ctcgcggagc cggcacagcg acaggcgccc
120
cg
122
<210> 19
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 19
gcggcggcgg gcggggagcc aggcccgagc tgcgttctgc gcagccattg gtgggcgccg
60
cgctctgcac tgagcatgtt cgcgccccgc cggcccctag ccgcagccgc agccgcagcg
120
ac
122
<210> 20
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 20
caaccggttc cgccgcgttt gtgggctggt agcccggaat acatttccca gaggccttcg
60
cggccgacgt gcttcgcgca ggaacgcagc cgcctcccga ctggaggacg cggtagcgga
120
gc
122
<210> 21
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 21
gctgcccgtg gtcaaactgg agtcgctgaa gcgctggaac gaagagcggg gcctctggtg
60
cgagaagggg gtgcaggtgc tgctgacgac ggtgggcgcc ttcgccgcct tcggcctcat
120
ga
122
<210> 22
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 22
cttcccggct ccccgcggtg cgcacccgct ggccactctg cgcacgcgcg ccgggtgccc
60
cggcctaagg ccgttgacct cgggttctcc ccggcacagt cgaatccacg ccagggccct
120
ca
122
<210> 23
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 23
gcgggggagg ttgcggggga ggctcggcgt ccccgctctc cgccccgcga caccgactgc
60
cgccgtggcc gccctcaaag ctcatggttg tgccgccgcc gccctcctgc cggcccggct
120
gg
122
<210> 24
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 24
tgtactactt cctctgccac ctggccttgg tagacgcggg cttcactact agcgtggtgc
60
cgccgctgct ggccaacctg cgcggaccag cgctctggct gccgcgcagc cactgcacgg
120
cc
122
<210> 25
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 25
aaaaaaaaaa aaaagcaatg agccgcaagc cttggactcg cagagctgcc ggtgcccgtc
60
cgagagcccc accagcgcgg ctcacgcctc agtctcgccg ccccaaggtg ggatccgacg
120
cc
122
<210> 26
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 26
cgagagggcc cggtccagca gcctctgggg cccagtgcgc agggcactgc gggccgattg
60
cgccccgggg ccaggaggcg ccgagaaagc aaaagcaaaa gccggcggcg ggtggaggtc
120
aa
122
<210> 27
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 27
cggccgcagt gtgccgcccg ctgcgctatg cggggctcgt ctccccgcgc ctatgtcgca
60
cgctggccag cgcctcctgg ctaagcggcc tcaccaactc ggttgcgcaa accgcgctcc
120
tg
122
<210> 28
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 28
cctggcgcga ccgccagcag cacccagcgc ggggccggga gctgctgggg gcccaggctc
60
cgctctcccc accgctctgc accgctgccg gctgcggaca gacccgatgc gccaccacca
120
cc
122
<210> 29
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 29
ccggagcgcg ctgctgccct ctaccggtca tccgtgcggc cggacaccgt gtcaggcccg
60
cgaggagggc tctgccgcag tcccggggaa cagcacccag cagcgccact gggagaggaa
120
ac
122
<210> 30
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 30
agtccagagc ggcgctgtgc agctggaagg gcgcgcgata gctcaagtta gaggcggccc
60
cggggcgcgg cgcaggacac aagacctcaa actggtactt gcacaggtag ccgttggcgc
120
gc
122
<210> 31
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 31
ggcggtgcga gctccccgcc tgcgggacgc acggagaccg cggtcagcgc gccgcctggc
60
cggcccagcg cgcccagccc gcgcccagcc ccgtccactc ccgtccagcc ccgccgcccg
120
gc
122
<210> 32
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 32
cggtagagtt tccaacacga aagcccgtgt ggtcgcgccg ggagctcacg gcgttccaag
60
cggcacttat cccgcgttga tgcccaggca ccccgcgcgc cctgtttcac caggcccagt
120
ca
122
<210> 33
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 33
ccagcggcag tagctgtagc agcttcagcg aagccggaga tgggcagaga gcgcgcgcgg
60
cgcagcagct ccagattcac tgctctcccc tgcagctccc cgcgcccccg ccgctgtcgc
120
tg
122
<210> 34
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 34
gtgttctctg cggcgggccg cgtccccgct gagcctcgcg gtgacagccg cctttggcag
60
cgagcgctcg gggcacttct atccccgcct ctcaaagggt ggggacagcc gtttccagat
120
tt
122
<210> 35
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 35
cggccgcgcc cccggcagcc cagggcgcgc ttccaccacg gtaccggtgg attcgccgtg
60
cgcagccgga agatggcgca gacgcacaaa gcacaccgat gctgcgccat gatagggccg
120
gc
122
<210> 36
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 36
tctcgcggcg caggcggcgg cggcagaggt ggggtcgcgc agcggaggca gctcgagctt
60
cgggatgcgc gctcgcttct tgggctcctc gctcgatctt actgccccct tttttctctc
120
cc
122
<210> 37
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 37
tcctccagcc agagtcggtg ggactggctg cgctgccctg aagtggttct ccaagcagcg
60
cggagggtgg cggacggcgg acggagccca ggggccgcgt cgggtgggga aacccgaact
120
cg
122
<210> 38
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 38
tgcgcatcgc tggctctggg ttccgccgaa tgcgtcctcc tggcggtgat ggctctggac
60
cgcgcggccg cagtgtgccg cccgctgcgc tatgcggggc tcgtctcccc gcgcctatgt
120
cg
122
<210> 39
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 39
aggggagctg cgaggcgaag tgttcttcag ggaagcgggc tcgagtctcc gcagctgcgg
60
cggcggcggc ggcgcgctgg gccggcggcg ggcgcgggca gggggccggg ggtgccgcgc
120
gg
122
<210> 40
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 40
cctccacccc cggggggttc ctgcgcactg aaagaccgtt ctccggcagg ttttgggatc
60
cggcgacggc tgaccgcgcg ccgcccccac gcccggttcc acgatgctgc aatacagaaa
120
gt
122
<210> 41
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 41
agagaggggt cccagaacga aggtggcggc acgagctctg cgctggcggc tgtggggggc
60
cggcgctcag gaccccaact ccatccaagt tgcgccgcgg tgggggcggg cggaggcggc
120
gc
122
<210> 42
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 42
aatctcccct cgggctcgac ggatgtgcgc cccagatgtg ctgacacatg tccgatgcct
60
cgctgccttg gaggtctccc cgctcgcgtg tctcttctct tcgcaccagc ggcggaaacc
120
gc
122
<210> 43
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 43
gctccgcttc tccgggtttt agcggaagcc tgcggggggc ggggtaaccg cggaagccgg
60
cggccgtggg cgcgcgggtt gggggctctc gcgccgctcc gggctctccc cccccccggc
120
tg
122
<210> 44
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 44
cgcgctccgc ttctccgggt tttagcggaa gcctgcgggg ggcggggtaa ccgcggaagc
60
cggcggccgt gggcgcgcgg gttgggggct ctcgcgccgc tccgggctct cccccccccc
120
gg
122
<210> 45
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 45
gcgagggatc tctgtgcgtc ctcactggcc catgcaccca gcacctgcga ctcccgccgt
60
cgggctgcgt ggccccgcgc ccacacctgc ccgtcccttc cgtcgtccct cgctcgcgca
120
ga
122
<210> 46
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 46
ggggaggtgt ggggagcgga aggccgcagg agcatctttg cggagaaagt actttggctg
60
cggcgggcgc agggcgggcc ggctagcccc gcgccccacc tgttctgtgc gtcgcgctcg
120
cc
122
<210> 47
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 47
tagggctgga aacccgccgc cacagcgggc tagaggtcgt ccccgcccgc aacatatgcg
60
cgaaggaaag tgctacgaac gtcaaatggc cgccccccgc cgacgccatc tgctctgcga
120
ag
122
<210> 48
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 48
cgcccgcaac atatgcgcga aggaaagtgc tacgaacgtc aaatggccgc cccccgccga
60
cgccatctgc tctgcgaagc agaaacggcg gcagctgcgc gcccagtccc tccgcccgcg
120
cc
122
<210> 49
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 49
ccccctgttc aaggtctgtc accgtagggg gcgggggggc gcgtggagcc gctgggggtt
60
cggcccaccc cgcgaaccga gctcccggcc ctgtgcgccc tcagctctgc cgcgggcgtt
120
gg
122
<210> 50
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 50
gctgtggccg cagctgaggc ccgacgagct tccggccggg tctttgccct tcactggccg
60
cgtgaacatc acggtgcgct gcacggtggc cacctctcga ctgctgctgc atagcctctt
120
cc
122
<210> 51
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 51
gtgtgcgtgt gcgtgtgctc agcctcagcg tgaggggcac ctgctcgtct gggctcacag
60
cgaaggcagc ctcgccgcga gctgccgctg ccgctgctgc cgccactggt gttgccgctc
120
tc
122
<210> 52
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 52
tggggcagcg gcgttgcagg agatgagctc agcgcaaagg gaaccccgca gcggcgagtg
60
cggctgctgg cctgcgcgct gtggccccaa caggctggca gggcgcgggc gggtggcggg
120
gt
122
<210> 53
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 53
agagtcggtg ggactggctg cgctgccctg aagtggttct ccaagcagcg cggagggtgg
60
cggacggcgg acggagccca ggggccgcgt cgggtgggga aacccgaact cgcggagggg
120
aa
122
<210> 54
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 54
aaagccctgg caggtaaaga gaggacccgc gcaggctggg agctcccact cctcctccag
60
cgtcacgctc gccctccgcc gctgcctcgc gtccgggtct gtttatatag cgtctggagg
120
cc
122
<210> 55
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 55
ctggccaagt gccggcccat cgcggtgcgc agcggagacg ccttccacga gatccggccg
60
cgcgccgagg tggccaacct cagcgcgcac agcgccagcc ccatccagga tgcggtcctg
120
aa
122
<210> 56
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 56
ggcgccggca gcttcgcgcc ggcggctgga agcgggcggg ctgcacgggc ggctcgagtg
60
cggggacccc agcccctcgc cctcgtgagc gccgcccctg ccacctgctg ccaagtcacc
120
gg
122
<210> 57
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 57
ccccggccgc gccgggcgcg gggctcggga ttcgggagac cgcgcggcgc cgaagccacg
60
cgtcagcccc actgtcccgc gcgcctcgcc ccaggcctcg ggctcttcct ccgcacctcg
120
ta
122
<210> 58
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 58
acgcggggac tggaaagggc gcctgggtgg gaagaggcgc tggcgggtga tcgtccccac
60
cgggccagtc cccgggatct gctgccgccc ctctccgaaa ttcacagcca gagcgggcgc
120
ac
122
<210> 59
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 59
tctgagaagt gtcctcctcg ctctcttata aaaacaggac ttgttgccga ggtcagcgcg
60
cgcatcgagt gtgccaggcg tgtgcgtggt ttctgctgtg tcattgcttt cacggaaggt
120
gg
122
<210> 60
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 60
gcgcccagac tgcgcgccgc gccgctgcgc ccaacattcc cgaggacggc ttcgcgggcg
60
cgtatcgtcc agaccggagc accgccccac cgctagcgca ggagacctgc cggggaagtc
120
gc
122
<210> 61
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 61
aaaggccgta ctctgccccc cgcgggaccc aggtccccgc ctgctgcaga gcgcactctg
60
cgcacgtcga gccgcgaaag gttcacagaa gaaaacaaga gaaagaagta gcaggcactg
120
ag
122
<210> 62
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 62
ggaatccatt cttttaagcc agggtttaaa actcttcaag caagtcatct gcaaaggtac
60
cgcttctacc attttaaaga taggattatg ttccctagga caactggatg agccctagga
120
ac
122
<210> 63
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 63
gaggagcgcg ccgctgcctc tggcgggctt tcggcttgag gggcaaggtg aagagcgcac
60
cggccgtggg gtttaccgag ctggatttgt atgttgcacc atgccttctt ggatcggggc
120
tg
122
<210> 64
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 64
ccctgtgcgt gccgccgcgc tgttgctcgc agtgtgctgg cgccgagctc ggtggacacg
60
cgcgcagtca gagctgcctc tcgccctcgc tagctgggct cgcagcctct tcctccctcc
120
ct
122
<210> 65
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 65
ctctttggca agtggtttgt gcatcaggag aaactttcca cctgcgagcc gaaccggcgc
60
cgagtgcgtg tgtttctgcc tttttttgtt gtcgttgcct ccacccctcc ccattcttct
120
ct
122
<210> 66
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 66
tggctgccag agcgagtgag gggcgcagag gcggcagaga gcggagagcc ccggtgtctc
60
cgcgagggcg gcggcggcca gcagacggcg atcgaggcgc gcgccacggc acggccagcg
120
ca
122
<210> 67
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 67
aagcgcgtgg agagccgaaa ggtgcggtgg gcgcagaggg cgggctggct gcggggcgac
60
cgcgcgccgg ggccatgccg cgctccttcc tggtggactc gctagtgctg cgcgaggcgg
120
gc
122
<210> 68
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 68
ggggtcgcca tgaccgagtg gcccaggccc gagcgaagcc cgcgcgcggt gagtccgccg
60
cggcccatcc gtccctccgc ccgccagagc gtccatcggg acgcccaccc gggagggtct
120
cg
122
<210> 69
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 69
ccgagcgctg cccccgccgg cccgcggctg ccagccggcc ctgcccgcgc ccgggccccg
60
cgagcggccg cacttcacct tacggagggg agataatgag atcaattaga ggcgccgtca
120
cc
122
<210> 70
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 70
ggcaaccctg actcggaccg ctcgggagag ccccaggaga ggccagcgcc gcgcagcagc
60
cgccccgctg cgcccacctc cccggctgct cccggagggc tcacaaaggc ggtggccgcc
120
cg
122
<210> 71
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 71
gcgggcggca gccgcaagcg aggaatccag cgcagggaaa gtagccccag tggggcccgg
60
cgcgtcagcc ccactcgcgt ggcaaaactt gcgggggccc ccgcgtgccg cgcctcagcc
120
ca
122
<210> 72
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 72
tcctcgccgt cggggtcctc ctcctctgcc gacgagttgt cactgggcga ggcgtagctg
60
cgctctacgc cgcggagggg cggcctcttg gaggcgggga ccgggtactc ccgctgcagc
120
cc
122
<210> 73
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 73
gctgctcgcg ctccgccgcc cgggagatgc ttcctcgcgc ggcgcagcgc tgaggccgtg
60
cgtgcgcccc ggctgcgctg cgcgctcccc acatacacaa gctctccatg tgagctgaca
120
gg
122
<210> 74
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 74
cttctcttga aaaggaggag aatcaacact gggctcacaa ctcatcagag ctgagtcata
60
cgtacatcag caggacctac gtgggaacca aatagcaaac tcaaattggg aaatttgagg
120
aa
122
<210> 75
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 75
ccgagagccc cgcctgcagg cggtgtagat acatgtagat actgtagata ctgtagatac
60
cgccccggcg ccgacttgat aaacggtttc gcctcttttg gaagccgcct gcgtgtccat
120
tt
122
<210> 76
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 76
tgaggagtga ggaggcagaa aggaccgaga acaaggggac ccggttccat ttctggaccc
60
cgtccgcagg ctgctcgccc gacttggggt cgctctgccc cggacgatca ggacagctgc
120
gt
122
<210> 77
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 77
caaatctata tgaaggatcg aattgcattg aactagcaaa cacacacaca cacacgcaca
60
cgcaaaaact gatgaaagct gaacaaggtc tgtagtctag tcaacagtac tgcactatgt
120
ga
122
<210> 78
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 78
acagtctctc gcctcaaaga tctccgccat tagtggtagc catttaagaa aacagaatta
60
cgatgaataa tgatttgaag ccaaaaagtc aaaatatctt atttcgcaac tgtaattgct
120
gg
122
<210> 79
<211> 122
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 79
ccacacaggc ctctccctcg gtgcggtagc gagggttgcg ggcccaaacg cccgcgccca
60
cggaggcgcc tgcgacgact agaagcttcc acagccatat gggggcaaag acggcccagt
120
ag
122
<210> 80
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 80
ggtttttggg taggaaggat agtag
25
<210> 81
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 81
aaacaaatct aacccctaaa aaaac
25
<210> 82
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 82
caaactaaac acactaaacc
20
<210> 83
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 83
gggtttttag ggttggtggt a
21
<210> 84
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 84
tcctcataat aataaataac aacc
24
<210> 85
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 85
tatgtatgtg gtggggtt
18
<210> 86
<211> 45
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 86
aatgatacgg cgaccaccga gatctacact ctttccctac acgac
45
<210> 87
<211> 52
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<220>
<221> AGTCATCG
<222> (25)..(32)
<400> 87
caagcagaag acggcatacg agatagtcat cggtgactgg agttcagacg tg
52
<210> 88
<211> 59
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<220>
<221> прочий_признак
<222> (34)..(38)
<223> n представляет собой a, c, g, или t
<400> 88
acactctttc cctacacgac gctcttccga tctnnnnngg gtttttaggg ttggtggta
59
<210> 89
<211> 58
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 89
gtgactggag ttcagacgtg tgctcttccg atcttcctca taataataaa taacaacc
58
<210> 90
<211> 67
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<220>
<221> прочий_признак
<222> (34)..(38)
<223> n представляет собой a, c, g, или t
<400> 90
acactctttc cctacacgac gctcttccga tctnnnnngg taggtttttg ggtaggaagg
60
atagtag
67
<210> 91
<211> 59
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: Синтетический
полинуклеотид
<400> 91
gtgactggag ttcagacgtg tgctcttccg atctaaacaa atctaacccc taaaaaaac
59
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| СОСТАВ, НАБОР И ПРИМЕНЕНИЕ ДЛЯ ВЫЯВЛЕНИЯ КОЛОРЕКТАЛЬНОГО РАКА | 2021 |
|
RU2836003C2 |
| ОБЪЕДИНЕННАЯ ОЧИСТКА И ИЗМЕРЕНИЕ МЕТИЛИРОВАНИЯ ДНК С СОВМЕСТНЫМ ИЗМЕРЕНИЕМ МУТАЦИЙ И/ИЛИ УРОВНЕЙ ЭКСПРЕССИИ мРНК В АВТОМАТИЧЕСКОМ РЕАКЦИОННОМ КАРТРИДЖЕ | 2017 |
|
RU2780660C2 |
| РЕАГЕНТ ДЛЯ ВЫЯВЛЕНИЯ, НАБОР ДЛЯ ВЫЯВЛЕНИЯ И СПОСОБ ВЫЯВЛЕНИЯ МЕТИЛИРОВАНИЯ ГЕНА ITGA4 | 2017 |
|
RU2739071C1 |
| Набор олигонуклеотидных праймеров и флуоресцентно-меченых зондов для выявления эпигенетических маркеров рака шейки матки | 2021 |
|
RU2760573C1 |
| КОГОРТЫ БИОМАРКЕРОВ МОЧИ, ПРОФИЛЬ ЭКСПРЕССИИ ГЕНОВ И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2014 |
|
RU2668164C2 |
| СПОСОБ IN VITRO ДИАГНОСТИКИ ИЛИ ПРОГНОЗИРОВАНИЯ КОЛОРЕКТАЛЬНОГО РАКА ИЛИ ЕГО ПРЕДРАКОВОЙ СТАДИИ | 2020 |
|
RU2831090C2 |
| КОНСТРУКТЫ Т-КЛЕТОЧНОГО РЕЦЕПТОРА И ИХ ПРИМЕНЕНИЕ | 2019 |
|
RU2785954C2 |
| РЕЖИМ ВВЕДЕНИЯ ДОЗ ДЛЯ ЛЕЧЕНИЯ ЗАБОЛЕВАНИЯ, МОДУЛИРУЕМОГО CSF-1R | 2021 |
|
RU2835316C1 |
| НЕОАНТИГЕННЫЕ КОМПОЗИЦИИ И ИХ ИСПОЛЬЗОВАНИЕ | 2020 |
|
RU2832574C2 |
| МОДИФИЦИРОВАННАЯ ДНК С ЗАМКНУТЫМИ КОНЦАМИ (зкДНК) | 2018 |
|
RU2800026C2 |

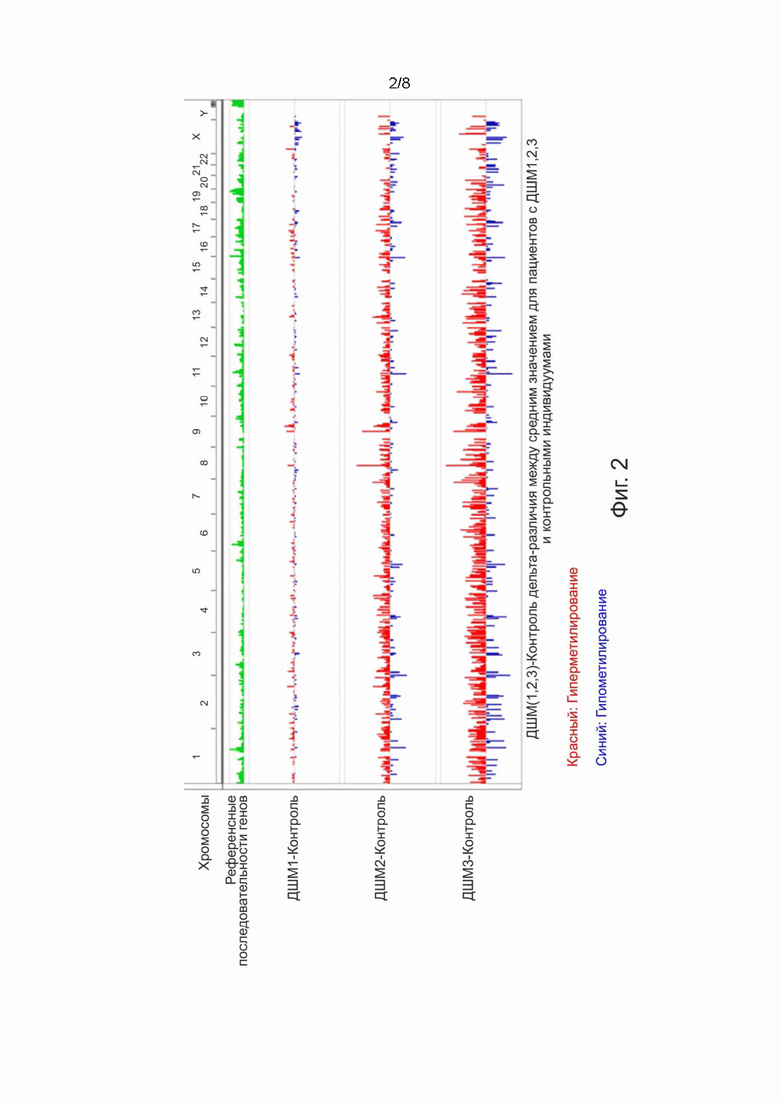
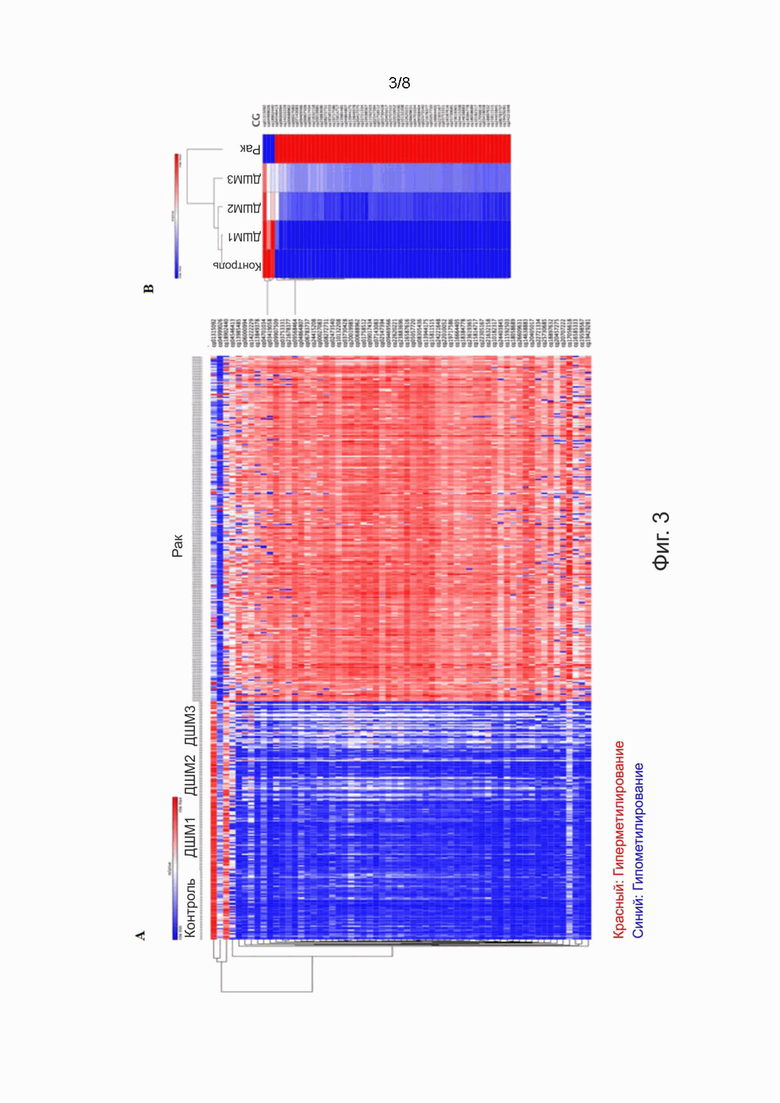
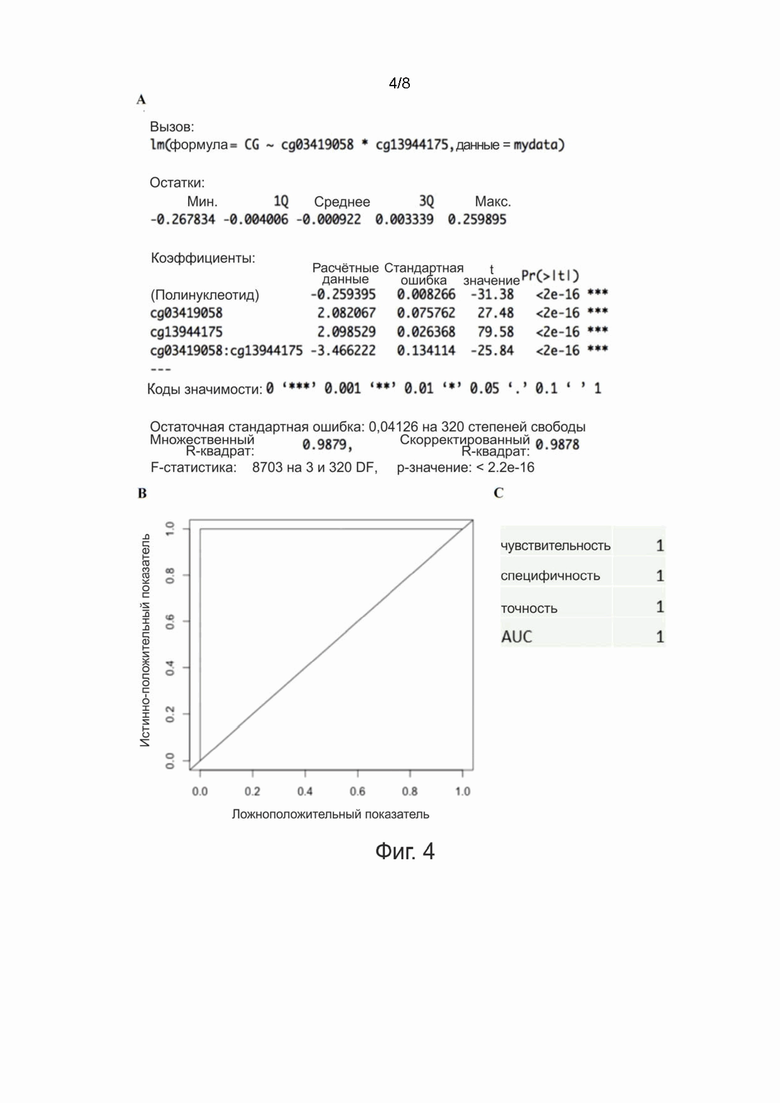
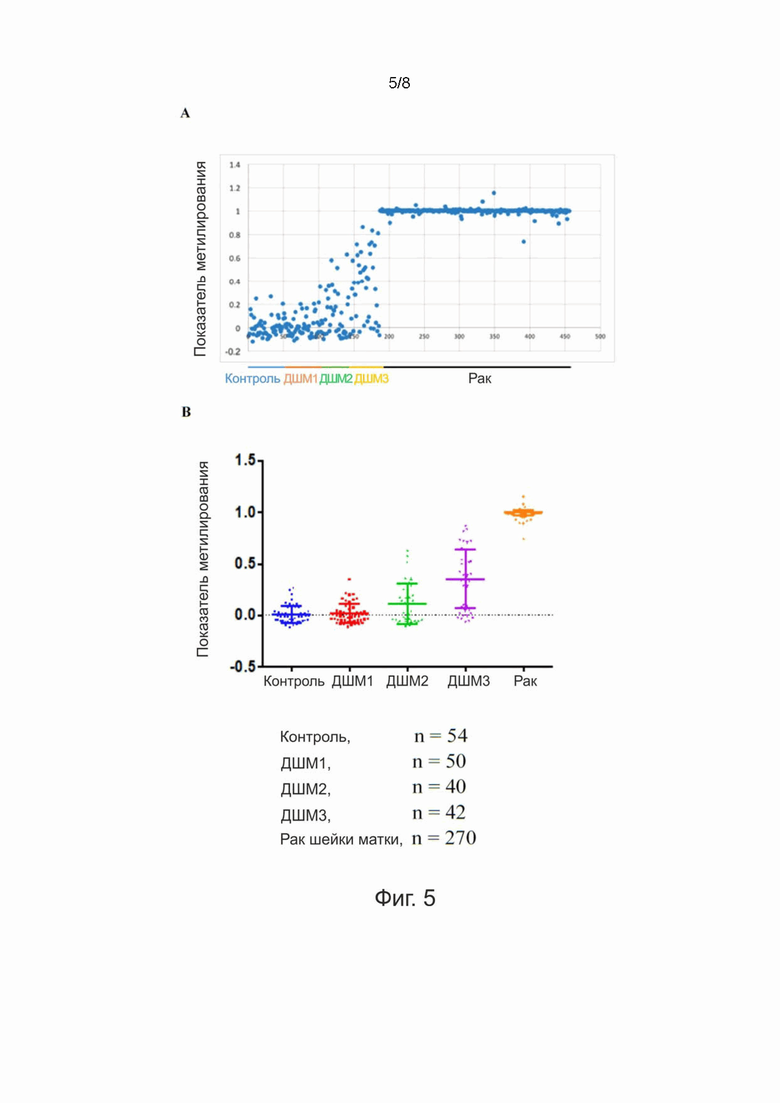
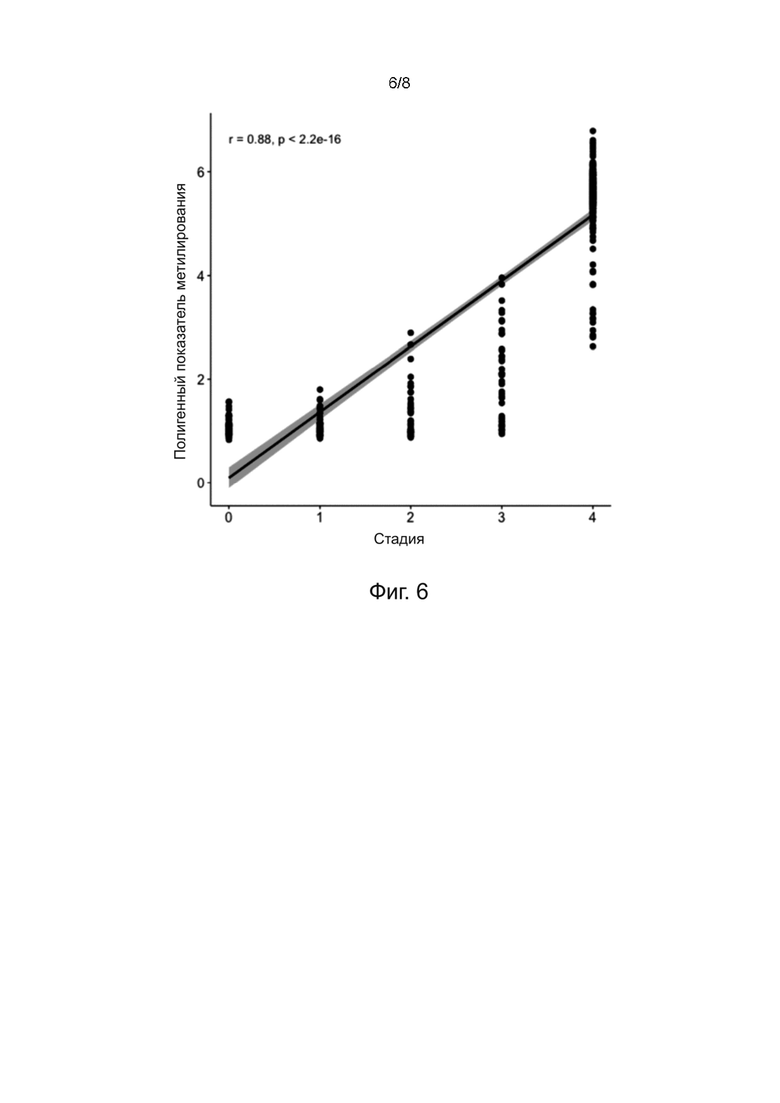
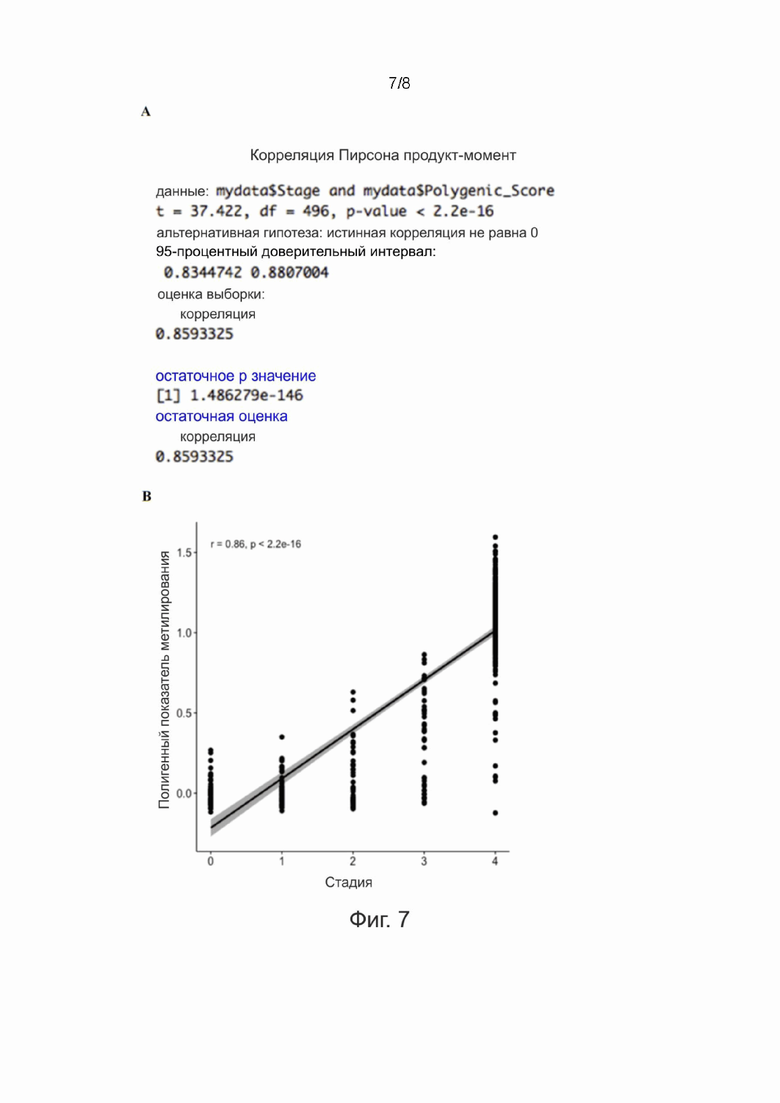
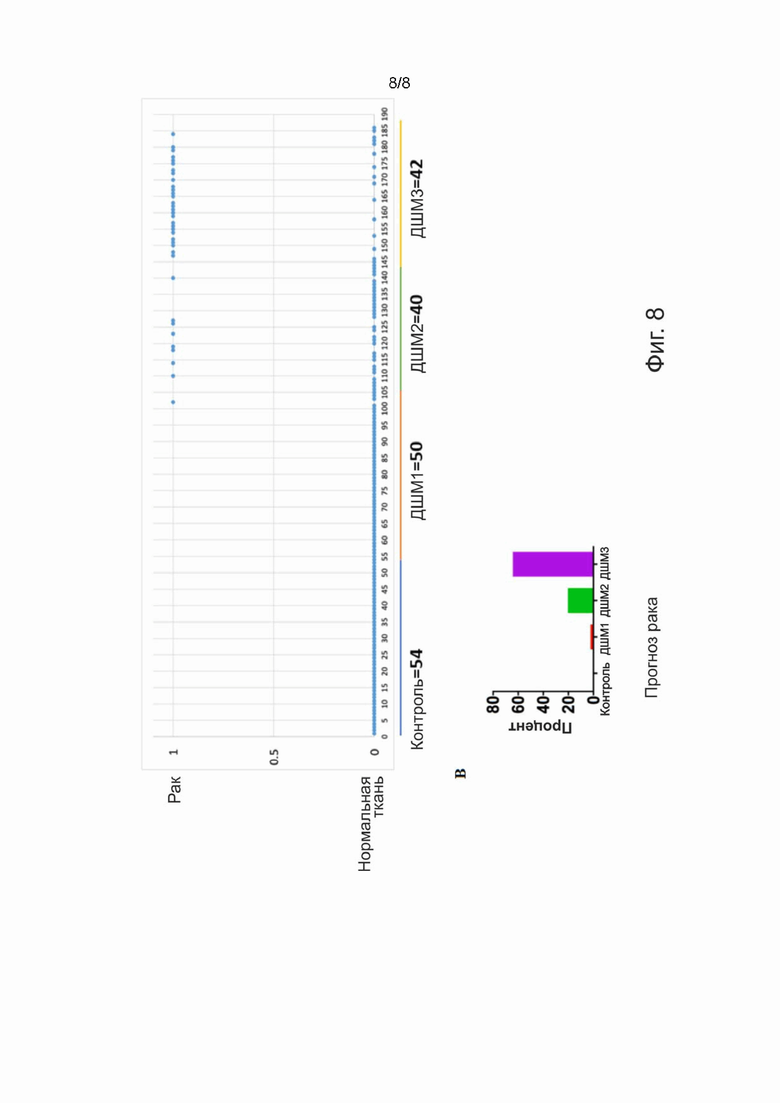
Изобретение относится к биотехнологии и биомедицине. Предложен набор для выявления рака шейки матки с использованием анализа метилирования CG-идентификаторов (CGID) с помощью бисульфитного секвенирования с мультиплексной целевой амплификацией. Указанный набор включает реагенты, необходимые для проведения указанного анализа CGID с SEQ ID NO: 3 и 31 и измерения уровней их метилирования. Изобретение позволяет расширить арсенал биомаркеров метилирования ДНК для раннего выявления рака шейки матки. 2 з.п. ф-лы, 8 ил., 3 табл., 7 пр.
1. Набор для выявления рака шейки матки с использованием анализа метилирования CG-идентификаторов (CGID) с помощью бисульфитного секвенирования с мультиплексной целевой амплификацией, включающий реагенты, необходимые для проведения указанного анализа CGID с SEQ ID NO: 3 и 31 и измерения уровней их метилирования.
2. Набор по п.1, где CGID, указанный в SEQ ID NO: 3, содержит праймеры, указанные в SEQ ID NO: 88 для прямого праймера и SEQ ID NO: 89 для обратного праймера.
3. Набор по п.1, где CGID, указанный в SEQ ID NO: 31, содержит праймеры, указанные в SEQ ID NO: 90 для прямого праймера и SEQ ID NO: 91 для обратного праймера.
| WO 2018158589 А1, 07.09.2018 | |||
| ESTELLER, M | |||
| Epigenetics in Cancer | |||
| N ENGL J MED | |||
| Станок для изготовления деревянных ниточных катушек из цилиндрических, снабженных осевым отверстием, заготовок | 1923 |
|
SU2008A1 |
| Зажим для канатной тяги | 1919 |
|
SU358A1 |
| WENTZENSEN, N | |||
| et al | |||
| Utility of methylation markers in cervical cancer early detection: appraisal of the state-of-the-science | |||
| GYNECOL ONCOL | |||
| Колосоуборка | 1923 |
|
SU2009A1 |
| FARKAS, S | |||
| A | |||
| et al | |||
| Genome-wide DNA methylation assay reveals | |||
Авторы
Даты
2024-04-16—Публикация
2020-02-04—Подача