Изобретение относится к рекомбинантной плазмидной ДНК рМВР- PUUV_N, обеспечивающей синтез и секрецию слитого белка MBP-PUUV_N в клетках Е. Coli, штамму Escherichia coli KRX/pMBP-PUUV_N, продуцирующему указанный белок и рекомбинантному белку PUUV_N, продуцируемому указанным штаммом и может быть использовано в биотехнологии, в частности в генной и белковой инженерии.
Вирус Пуумала (PUUV) относится к роду Orthohantavirus семейства Hantaviridae порядка Bunyavirales. Является возбудителем геморрагической лихорадки с почечным синдромом (ГЛПС). ГЛПС - острая вирусная зооантропонозная инфекция, вызываемая представителями рода Orthohantavirus, широко представленными по всему миру. Заболевание характеризуется лихорадкой и гриппоподобными симптомами, острой почечной недостаточностью и геморрагической гемостазиопатией в тяжелых случаях. На территории Российской Федерации (РФ) ежегодно регистрируется 7-9 тысяч случаев ГЛПС, возбудителем более 95% из них является PUUV. PUUV представляет собой оболочечный вирус с сегментированным одноцепочечным РНК-геномом с отрицательным смыслом. Люди инфицируются в большинстве случаев после прямого контакта с инфицированными грызунами или их экскрементами, зачастую при вдыхании аэрозоля, содержащего вирус. Геном вируса имеет три сегмента. Большой L-сегмент кодирует вирусную РНК-зависимую РНК-полимеразу (RdRp), средний М-сегмент кодирует два поверхностных гликопротеина (Gn) и (Gc), и малый S-сегмент кодирует белок нуклеокапсида (N).
На сегодняшний день в мире существует ряд вакцинных препаратов, направленных на специфическую профилактику ГЛПС. Все эти препараты относятся к классу инактивированных вакцин, их использование одобрено в Китае, Южной и Северной Корее. Препараты, производимые в этих странах разработаны на основе вирусов Хантаан и Сеул, и не способствуют формированию протективного иммунного ответа против PUUV. Работа с представителями рода Orthohantavirus и производство хантавирусных инактивированных вакцин сопряжено с созданием лабораторий и производств высокого уровня биобезопасности BSL-3, что значительно снижает, исследовательские возможности и возможности технологического масштабирования производства вакцинных препаратов.
На данный момент актуальной остаётся задача создания вакцинных препаратов против возбудителей ГЛПС распространённых на территории РФ. Для изготовления и контроля вакцинных препаратов против PUUV необходимо решить проблему разработки эффективных серологических методов на основе гликопротеинов Gn, Gc и нуклеопротеина N, участвующих в иммунореакции. Однако, до настоящего времени задача получения полноразмерных коммерческих препаратов белков Gn, Gc хантавирусов не решена, что обусловлено их токсичностью по отношению к бактериальным клеткам. В то же время, установлено, что нуклеопротеин N является иммунодоминантным антигеном.
Известно решение по патенту (RU2221869C2, МПК C12N 15/63, опубл. 20.10.2004) [1], где был предложен вариант рекомбинантной плазмиды с промотором SP-6, обеспечивающий синтез РНК-копии S-сегмента вируса Хантаан для положительного контроля для диагностики возбудителя геморрагической лихорадки с почечным синдромом. Получение вирусной РНК достигалось трансформацией Е. Coli штамма XL-2Blue плазмидой pSP64-S, содержащей кДНК-копию S-сегмента вируса Хантаан.
Известно решение по патенту (RU2495938C2, МПК С07К14/175, опубл. 10.07.2013) [2], где был предложен способ получения рекомбинантного антигена G2 хантавируса Добрава в клетках Е. coli.
Наиболее близким аналогом (прототипом) является изобретение (WO1998014585A1, МПК С07К14/175, опубл. 09.04.1998 г.) [3], которое относится к гену нуклеокапсида штамма R22 SEOUL_N (нуклеопротеина N) вируса Сеул, рекомбинантному вектору экспрессии указанного гена, трансформированному микроорганизму E.coli указанной рекомбинантной плазмидой и белку нуклеокапсида, экспрессируемому указанным трансформантом, а также к новой фармацевтической композиции для диагностики или профилактикЬ геморрагической лихорадки с почечным синдромом, вызванной вирусами рода Hantavirus, содержащими указанный белок нуклеокапсида. Белок SEOUL_N нарабатывали в тельцах включения для получения растворимого белка. При очистке проводили денатурацию и последующую ренатурацию указанного белка.
Однако белки, приведенные в аналогах и прототипе не способствуют формированию протективного иммунного ответа против PUUV.
Таким образом, из анализа уровня техники видно, что существует потребность в разработке технологии и средств для получения рекомбинантного белка PUUV_N.
Раскрытие изобретения
Техническим результатом изобретения является синтез белка МВР- PUUV_N, из которого получают очищенный рекомбинантный белок PUUV_N с чистотой препарата не менее 95% для диагностики ГЛПС.
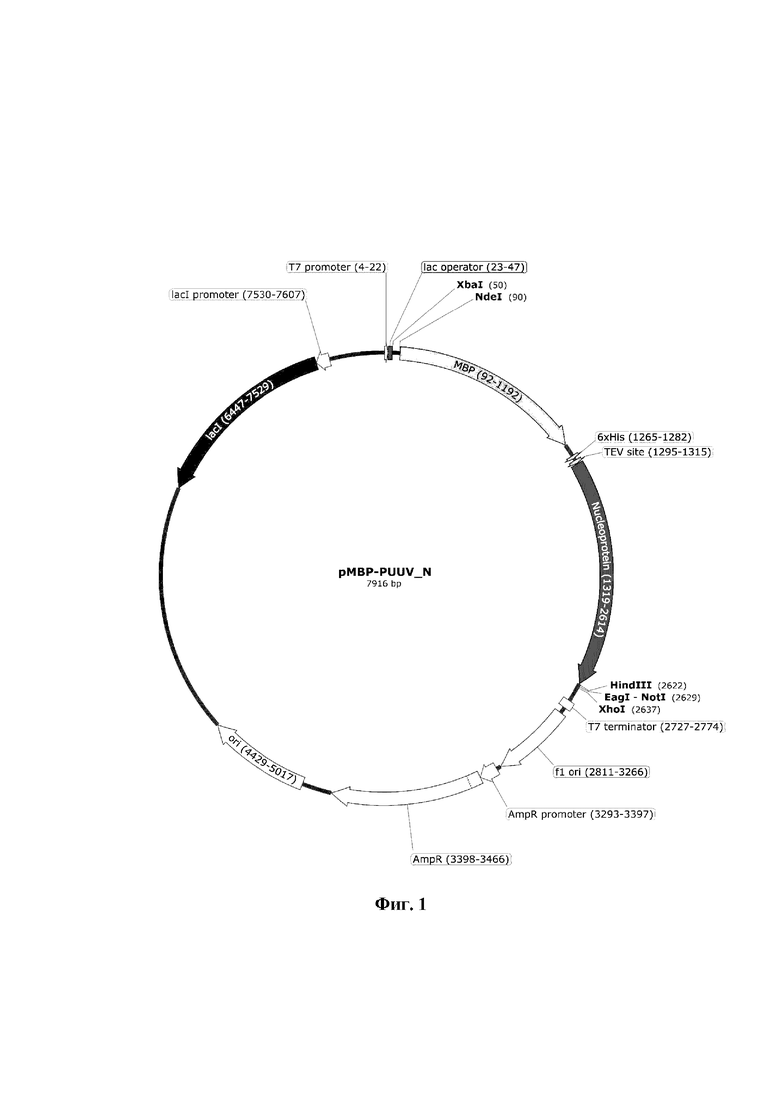
Указанный технический результат достигается тем, что получена рекомбинантная плазмидная ДНК pMBP-PUUV_N, обеспечивающая синтез и секрецию слитого рекомбинантного белка MBP-PUUV_N в клетках Е. coli, имеющая размер 7916 п.н. и содержащая следующие элементы генетической конструкции в соответствии с физической и генетической картой, представленной на фиг. 1:
- ген, кодирующий сигнальную аминокислотную последовательность мальтоза-связывающего белка (МВР), имеющий координаты с 92 по 1192 п.н.;
- в открытой рамке считывания с МВР ген рекомбинантного нуклеопротеина вируса Пуумала (PUUV_N), с нуклеотидной последовательностью SEQ ID NO: 1, имеющий координаты с 1319 по 2614 п.н.;
- нуклеотидную последовательность в рамке считывания гена МВР, кодирующую аминокислотные остатки 6-ти гистидинов (б-His), имеющий координаты с 1265 по 1282 п.н.;
- нуклеотидную последовательность в рамке считывания гена МВР сайта узнавания и разрезания TEV-протеазой с 1295 по 1315 п.н.;
- промотор и терминатор транскрипции Т7-РНК-полимеразы, имеющие координаты с 4 по 22 п.н. и с 2727 по 2774 соответственно;
- ген Р-лактамазы (AmpR), в качестве генетического маркера, определяющего устойчивость к пенициллиновым антибиотикам клеток бактерии Е. Coli, имеющий координаты с 3398 по 3466 п.н.;
- уникальные сайты рестрикции Xbal, Ndel, Hindlll, Xhol, имеющие координаты 50, 90, 2622, 2637, соответственно;
- lacl, имеющий координаты с 6447 по 7529 п.н.;
- lacl промотор, имеющий координаты с 7530 по 7607 п.н.;
- ori, имеющий координаты с 4429 по 5017 п.н.;
- fl ori, имеющий координаты с 2811 по 3266 п.н.;
- сайт рестрикции Eagl-Notl, имеющий координаты 2629 п.н.;
- ac оператор, имеющий координаты с 23 по 47 п.н.;
- AmpR промотор, имеющий координаты с 3293 по 3397 п.н.
Указанный технический результат также достигается созданием рекомбинантного штамма Escherichia coli KRX/pMBP-PUUV_N, полученного путем трансформации клеток штамма Escherichia coli KRX рекомбинантной плазмидной ДНК pMBP-PUUV_N по п. 1., продуцирующий гибридный полипептид MBP-PUUV_N, используемый для получения рекомбинантного белка PUUV_N, применяемого для диагностики геморрагической лихорадки с почечным синдромом.
Существенными отличиями заявляемого изобретения от прототипа, обеспечивающими достижение технического результата, являются:
1. Использование нуклеопротеина вируса Пуумала вместо вируса Сеул.
2. Использование в качестве белка слияния пептида МВР, обеспечивающего биосинтез PUUV_N в растворимой форме, что позволяет избежать этапа денатурации и последующей ренатурации рекомбинантного белка.
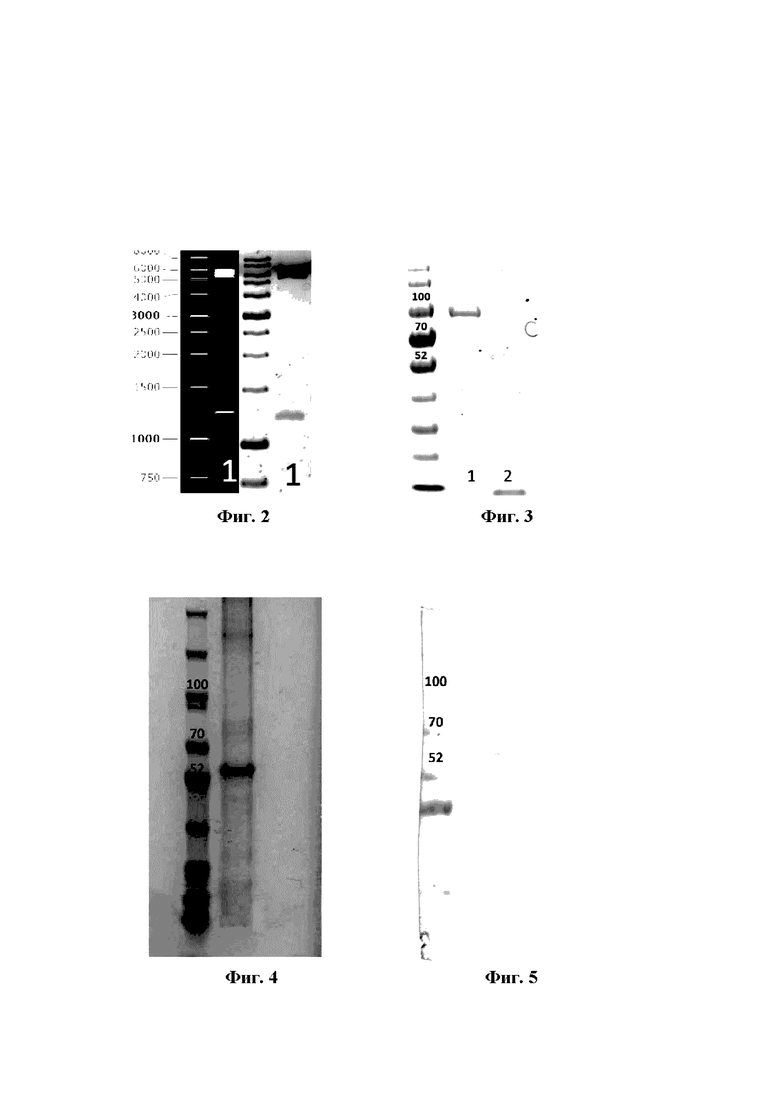
Сущность заявленного изобретения поясняется чертежами. На фиг. 1 изображена физическая и генетическая карта рекомбинантной плазмиды pMBP-PUUV_N. На фиг. 2 представлен рестрикционный анализ экспрессионной конструкции pMBP-PUUV_N эндонуклеазами рестрикции ВатШ и Xhol. Слева предполагаемая электрофореграмма. На фиг. 3 изображена электрофореграмма лизата клеток штамма-продуцента Е. coli KRX/pMBP-PUUV_N. 1 - лизат клеток штамма-продуцента Е. coli KRX/pMBP-PUUV_N, 2 - лизат клеток Е. coli KRX.
На фиг. 4 приведена электрофореграмма препарата рекомбинантного белка PUUV_N. На фиг. 5 представлен Вестерн-блот рекомбинантного белка PUUV_N с использованием моноклональных антител 5F147 (MyBioSource, США) в разведении 1:500. На фиг. 6 представлены: SEQ ID N0:1 - нуклеотидная последовательность гена рекомбинантного нуклеопротеина вируса Пуумала (PUUV_N), кодирующая полипептид MBP-PUUV_N и SEQ ID NO: 2 - аминокислотная последовательность рекомбинантного белка PUUV N.
Осуществление изобретения.
Пример 1. Конструирование рекомбинантной плазмидной ДНК pMBP-PUUV_N, предназначенной для экспрессии слитого рекомбинантного белка MBP-PUUV_N.
Фрагмент ДНК, кодирующий белок нуклеопротеина PUUV_N, получили амплификацией с генетической конструкции, содержащей полноразмерную кДНК копию S-сегмента. Использовали праймеры, фланкирующие открытую рамку считывания белка PUUV_N и вносящие на 5’-конец последовательности сайт узнавания эндонуклеазой рестрикции BamHI и сайт узнавания эндонуклеазой рестрикции XhoI на 3’-конец фрагмента. ДНК фрагмент, кодирующий белок PUUV_N, лигировали с плазмидой pMBP предварительно обработанной эндонуклеазами рестрикции BamHI и XhoI. Генетические конструкции трансформировали в химически компетентные клетки E. Coli NEB Stable (NEB, США). Спустя 16 часов культивирования на агаризированой среде LB (100 мкг/мл ампициллина) при температуре 30°C, отбирали единичные колонии и культивировали 12 часов в жидкой питательной среде LB с селективным антибиотиком (100 мкг/мл ампициллина) при температуре 30°C. Плазмидную ДНК экстрагировали с использованием набора QIAprep Spin Miniprep (Qiagen, Германия). Корректность генетических конструкций подтверждали рестрикционным анализом по сайтам узнавания эндонуклеазами рестрикции BamHI и XhoI фиг. 2 и секвенированием по методу Сенгера.
Пример 2. Получение штамм-продуцента путем трансформации плазмидной ДНК pMBP-PUUV_N клеток Escherichia coli KRX.
Штамм-продуцент рекомбинантного белка MBP-PUUV_N получали электропорацией компетентных клеток Escherichia coli KRX (Promega, США). Трансформанты культивировали на агаризированой среде SOB, содержащей 1% глюкозы и 100 мкг/мл ампициллина. Полученную культуру засевали в жидкую среду SOB, содержащую 100 мкг/мл ампициллина, культивировали 6-8 ч при 37°С в термостатируемом орбитальном шейкере при 220 об/мин до достижения OD600 ~ 1.
Пример 3. Биосинтез рекомбинантного слитого белка MBP-PUUV_N путём индуцированного культивирования штамм-продуцента Escherichia coli KRX/ pMBP-PUUV_N.
Продукцию рекомбинантного белка индуцировали добавление L-рамнозы до концентрации 0,1% и добавлением изопропил-β-D-1-тиогалактопиранозида (ИПТГ) до концентрации 1 мМ, индукцию экспрессии проводили в течение 20-24 ч при 18°С при 220 об/мин.
Пример 4. Выделение, отщепление белка слияния и очистка рекомбинантного белка PUUV_N из клеток Escherichia coli KRX/ pMBP-PUUV_N.
Бактериальную биомассу осаждали посредством центрифугирования при 4500 g в течение 15 мин при 4°С, осадок ресуспендировали в фосфатном буфере А (20 мМ NaH2PO4, 500 мМ NaCl, 25 мМ имидазол, pH 8.0) 15 мл буфера на 1 грамм клеточного осадка. Лизис клеток осуществляли гомогенизацией под давлением в гомогенизаторе EmulsiFlex-C3 (Avestin, Канада) (фиг. 3). Лизат центрифугировали при 13000 g в течение 30 мин при 4°С. Выделение рекомбинантных белков из супернатанта осуществляли методом металл-хелатной аффинной хроматографии. Супернатант наносили на сорбент Sepharose Ni-NTA (GE Healthcare, Великобритания). После колонку промывали фосфатным буфером А (20 мМ NaH2PO4, 500 мМ NaCl, 25 мМ имидазол pH 8.0) со ступенчатым повышением концентрации имидазола: 50, 100 и 150 мМ. Целевые белки элюировали фосфатным буфером Б (20 мМ NaH2PO4, 500 мМ NaCl, 500 мМ имидазол, pH 8.0). Полученный хроматографически очищенный белок диализовали против буфера А (20 мМ NaH2PO4, 500 мМ NaCl, 25 мМ имидазол, pH 8.0,) и обрабатывали протеазой TEV (Invitrogen, США) в массовом соотношении протеазы к белку 1:1000, при температуре 4°С в течение 16 ч для отщепления последовательности пептида MBP от целевого белка. Продукты расщепления разделяли с использованием металл-хелатной аффинной проточной хроматографии. Белковую фракцию наносили на колонку, содержащую Sepharose Ni-NTA, пептид MBP, TEV протеаза и балластные белки связывались с сорбентом, целевой белок оставался несвязанным (фиг. 4). Белковые фракции, собранные в процессе очистки, разделяли в 10% ПААГ в денатурирующих условиях по методике Лэммли. Таким образом, получена система для наработки рекомбинантного PUUV_N. Проведение двух последовательных раундов метал-хелатной аффинной хроматографии позволяет получить высокоочищенные препарат рекомбинантных вирусных белков.
Пример 5. Определение антигенной активности рекомбинантного белка PUUV_N в Вестерн-блотинге.
Выявление антигенной активности препарата рекомбинантного белка PUUV_N проводили с использованием метода Вестерн-блот анализа. Пробы из фракций, собранных в процессе очистки, разделяли в 10% ПААГ в денатурирующих условиях по методике Лэммли. Из геля белок переносили на нитроцеллюлозную мембрану. Затем мембрану помещали в блокирующий буфер (10 mM Трис-HCl, 150 mM NaCl, 0.05% Твин-20, 3% рыбий желатин, pH 7.5) на 1 ч при 37°С. Далее мембрану инкубировали с использованием моноклонального антитела 5F147 (MyBioSource, США) в разведении 1:500, в течение 1 ч при 37°С. После 3-кратной отмывки в буфере ФБРТ (10 mM Трис-HCl, 150 mM NaCl, 0.05% Твин-20, pH 7.5) мембрану инкубировали при комнатной температуре в течение 1 часа в разведении 1:10000 с пероксидазным конъюгатом антител кролика к IgG мыши. После 3-кратной отмывки проводили визуализацию образовавшихся иммунных комплексов добавлением 0,3% раствора диаминобензидина в 0,1% перекиси водорода (фиг. 5).
Пример 6. Сравнение антигенной активности рекомбинантного белка PUUV_N c нативным антигеном в иммуноферментном анализе.
Препарат нативного антигена получен в результате инфицирования чувствительной клеточной культуры (Vero) вирусом Пуумала. Через 5 суток выявлена 100 % деградация монослоя клеток в результате цитопатического действия. Детрит отделили от культуральной жидкости центрифугированием. Культуральную жидкость дезинфицировали добавлением 0,1% пропиолактона. Проверили отсутствие остаточной инфекционности тремя пассажами аликвоты на чувствительной культуре клеток. Использовали в качестве антигена для иммуносорбента в ИФА. Антигены рекомбинантного белка N и нативный развели 1:100 в растворе 0,1М натрия углекислого и внесли по 100 мкл в лунки планшета для иммунологических реакций. Инкубировали 18 часов при температуре 21°С, аспирировали жидкость, блокировали сайты связывания полистирола раствором 0,1% казеина по 200 мкл в лунку в течении 2 часов при температуре 21°С. Жидкость аспирировали, планшет высушивали 18 часов при температуре 21°С. Сравнительный анализ проведен с использованием коллекции из 35 образцов сыворотки крови пациентов медицинских учреждений с диагнозом хантавирусная инфекция в результате инфицирования вирусом Пуумала. Их специфическая активность была определена при заборе крови в титрах реакции непрямой иммунофлуоресценции (РНИФ). Образцы разводили от 1:100 до 1:102400 серией последовательных двукратных разведений в растворе 0,1% бычьего сывороточного альбумина и вносили по 100 мкл разведений в дубле в лунки обоих планшетов с рекомбинантным и нативным антигеном. В качестве отрицательного контроля исследовали 4 образа от условно здоровых доноров методом скрининга в разведении 1:100. Инкубировали 60 мин при температуре 37°С. Содержимое лунок аспирировали и пятикратно отмывали не связавшиеся в иммунные комплексы реагенты фосфатно-солевым раствором с добавление 0,2% Твин-20 (ФСБ-Т). Вносили в лунки по 100 мкл раствора пероксидазного конъюгата МАТ мыши к IgG человека 1:100 в 0,1% казеине, инкубировали 30 мин при температуре 37°С, аспирировали из лунок жидкость и пятикратно отмывали ФСБ-Т. Вносили по 100 мкл хромогена тетраметилбензидина (ТМБ) с визуализацией образовавшихся иммунных комплексов 15 мин при 37°С. Останавливали реакцию внесением 1М раствора серной кислоты, результаты учитывали на планшетном ридере при длине волны 450 нм. Титром образца считали максимальное разведение (в повторе), показавшее значение оптической плотности в четыре раза превышающее среднеарифметическое значение заведомо отрицательных образцов. В результате выявления титров специфических антител к антигенам рекомбинантного белка N и нативного вируса Пуумала в обоих случаях 35 клинических образцов показали положительный результат более 1:100 (табл. 1). В таблице 1 дан сравнительный анализ титров специфических к вирусу Пуумала антител в образцах сыворотки крови пациентов. Ложно положительных результатов при исследовании образцов от условно здоровых доноров не выявлено. Ранговый коэффициент корреляции Спирмена в парах данных «РНИФ-ИФА N» составил 0,37. Аналогичная величина в парах «ИФА N - ИФА нат» равна 0,91. Специфические IgG в крови инфицированных пациентов выявлены титрах от 1:200 до 1:102400. Ранговый коэффициент корреляции Спирмена в парах данных «РНИФ-ИФА N» свидетельствует о слабо положительной корреляции, тогда как аналогичная величина в парах «ИФА N-ИФА нат» говорит о высокой корреляции результатов. Таким образом, каждый из антигенов продемонстрировал способность выявлять специфические IgG в крови инфицированных пациентов в сравнимых титрах ИФА при отсутствии ложно положительных результатов.
Таблица 1. Сравнительный анализ титров специфических к вирусу Пуумала антител в образцах сыворотки крови пациентов
По результатам исследования 35 образцов с IgG к Пуумала и 4 образца без специфических антител, применение в ИФА рекомбинантного белка N как антигена может обеспечить выявление специфических IgG к вирусу Пуумала на уровне требований к диагностическим характеристикам (чувствительность, специфичность) современных медицинских изделий.
Таким образом, получена система для наработки и очистки рекомбинантного белка PUUV_N. Результаты иммунологических исследований продемонстрировали возможность использования данных препаратов для серологических исследований.
ПРИЛОЖЕНИЕ
Перечень последовательностей, представленная в соответствии со стандартом ST26 ВОИС
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3"
fileName="Приложение_Рекомбинантный белок PUUV_N.xml"
softwareName="WIPO Sequence" softwareVersion="2.3.0"
productionDate="2023-10-27">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>123456</ApplicationNumberText>
<FilingDate>2023-10-19</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>12345</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1234567</ApplicationNumberText>
<FilingDate>2023-10-06</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное бюджетное учреждение
науки «Государственный научный центр вирусологии и биотехнологии
«Вектор» Федеральной службы по надзору в сфере защиты прав
потребителей и благополучия человека (ФБУН ГНЦ ВБ «Вектор»
Роспотребнадзора)</ApplicantName>
<ApplicantNameLatin>Federalnoe byudzhetnoe uchrezhdenie nauki
"Gosudarstvennyj nauchnyj tsentr virusologii i biotekhnologii
"Vektor" Federalnoj sluzhby po nadzoru v sfere zashchity
prav potrebitelej i blagopoluchiya cheloveka (FBUN GNTS VB
"Vektor" Rospotrebnadzora) (RU)</ApplicantNameLatin>
<InventionTitle languageCode="ru">Рекомбинантная плазмидная ДНК
pMBP-PUUV_N, обеспечивающая синтез и секрецию слитого белка
MBP-PUUV_N в клетках E. Coli, штамм Escherichia coli
KRX/pMBP-PUUV_N, продуцирующий указанный белок и рекомбинантный
белок PUUV_N, продуцируемый указанным штаммом и используемый для
диагностики геморрагической лихорадки с почечным
синдромом</InventionTitle>
<SequenceTotalQuantity>2</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>1299</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1299</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tccgacttgacagacatccaagaggagataacccgccatgagcaacaac
ttgttgttgccagacaaaaactcaaggatgcagagagagcagtggaagtggacccggatgacgttaacaa
aaacacactacaagcgaggcaacaaacagtgtcagcattggaggataaactcgcagactacaagagaaga
atggcagatgctgtgtcccggaagaaaatggatactaaacctactgacccgactgggattgaacctgatg
atcatctcaaggagagatcaagccttagatatggaaatgtccttgatgtaaatgccattgacattgaaga
accaagtggtcagacagcagattggtatactattggagtctatgtaataggttttacaattcccatcatt
ttaaaggctctatacatgttgtcaacacgtggaagacagaccgtgaaagagaacaagggaacacggatca
ggttcaaggatgacacatcatttgaggatattaatggcattaggagaccaaaacatctatatgtgtccat
gcctactgcccagtccaccatgaaagctgaagaacttacacccggacggtttcgtacaattgtatgtggc
ttattccctacacagatacaagtgcgtaatatcatgagtccagtgatgggagtgattggtttttcttttt
ttgtcaaagactggccagagaggattagggaatttatggaaaaagaatgccctttcataaaaccagaagt
taagcccgggacaccagcacaggagatagaatttttgaaaaggaatagggtttatttcatgacccgccaa
gatgttcttgacaaaaatcatgtggctgacattgataagttaattgactatgctgcctctggtgacccta
catcacctgatgacatcgaatcgcctaatgcaccatgggtatttgcttgtgcaccagaccggtgtccccc
tacatgcatttatgttgctgggatggctgaattaggcgcattcttttcaatcctacaggatatgaggaac
actattatggcatctaaaactgtaggtacagcagaagagaaattaaaaaagaagtcctccttctatcaat
catatttacgccggacacaatcaatggggattcaacttgatcagaggataatcctactgtacatgttgga
gtggggaaaagaaatggtggatcatttccatcttggtgatgatatggatcctgagctcaggggccttgct
cagtcactcatagatcagaaagtaaaagaaatatcaaatcaggagcccttaaagatatga</INSDSeq_
sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>432</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..432</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SDLTDIQEEITRHEQQLVVARQKLKDAERAVEVDPDDVNKNTLQARQQT
VSALEDKLADYKRRMADAVSRKKMDTKPTDPTGIEPDDHLKERSSLRYGNVLDVNAIDIEEPSGQTADWY
TIGVYVIGFTIPIILKALYMLSTRGRQTVKENKGTRIRFKDDTSFEDINGIRRPKHLYVSMPTAQSTMKA
EELTPGRFRTIVCGLFPTQIQVRNIMSPVMGVIGFSFFVKDWPERIREFMEKECPFIKPEVKPGTPAQEI
EFLKRNRVYFMTRQDVLDKNHVADIDKLIDYAASGDPTSPDDIESPNAPWVFACAPDRCPPTCIYVAGMA
ELGAFFSILQDMRNTIMASKTVGTAEEKLKKKSSFYQSYLRRTQSMGIQLDQRIILLYMLEWGKEMVDHF
HLGDDMDPELRGLAQSLIDQKVKEISNQEPLKI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Минигеномная система вируса Пуумала для оценки противовирусной активности ингибиторов РНК-зависимой РНК-полимеразы | 2022 |
|
RU2783430C1 |
| Рекомбинантная плазмидная ДНК pET21-NPVE, содержащая ген нуклеопротеина (NP) вируса Эбола и рекомбинантный белок NP-ВЭ, полученный в результате экспрессии гена NP вируса Эбола с использованием рекомбинантной плазмидной ДНК pET21-NPVE и обладающий иммуногенными и антигенными свойствами | 2020 |
|
RU2739505C1 |
| Рекомбинантная плазмида pET21-GST-ND, обеспечивающая синтез и секрецию эктодомена Gn гликопротеина вируса Хантаан, и рекомбинантный штамм Escherichia coli BL21(DE3)pLysE pET21-GST-ND - продуцент белка Gn - эктодомена гликопротеина вируса Хантаан | 2023 |
|
RU2809199C1 |
| Рекомбинантная плазмида pET21-GST-CD, обеспечивающая синтез и секрецию эктодомена Gc гликопротеина вируса Хантаан, и рекомбинантный штамм Escherichia coli BL21(DE3)pLysE pET21-GST-CD - продуцент белка Gc - эктодомена гликопротеина вируса Хантаан | 2023 |
|
RU2807520C1 |
| Пептидные иммуногены и вакцинная композиция против коронавирусной инфекции COVID-19 с использованием пептидных иммуногенов | 2020 |
|
RU2743593C1 |
| Рекомбинантная плазмидная ДНК pET21-VP40VE, содержащая ген матриксного белка VP40 вируса Эбола и рекомбинантный белок VP40-ВЭ, полученный в результате экспрессии гена белка VP40 вируса Эбола с использованием рекомбинантной плазмидной ДНК pET21-VP40VE и обладающий иммуногенными и антигенными свойствами | 2020 |
|
RU2742511C1 |
| Пептидные иммуногены, используемые в качестве компонентов вакцинной композиции против коронавирусной инфекции COVID-19 | 2020 |
|
RU2743594C1 |
| ШТАММ БАКТЕРИЙ ESCHERICHIA COLI KRX pET32b/ASFV/p30-ПРОДУЦЕНТ ХИМЕРНОГО РЕКОМБИНАНТНОГО БЕЛКА p30 ВИРУСА АФРИКАНСКОЙ ЧУМЫ СВИНЕЙ | 2017 |
|
RU2647573C1 |
| Штамм бактерий Escherichia coli - продуцент рекомбинантного белка NS1 | 2021 |
|
RU2789735C1 |
| Штамм бактерий Escherichia Coli - продуцент рекомбинантного белка IL-29 | 2020 |
|
RU2760585C1 |

Изобретение относится к биотехнологии. Предложена рекомбинантная плазмидная ДНК pMBP-PUUV_N, обеспечивающая синтез и секрецию слитого рекомбинантного белка МВР-PUUV_N в клетках Е. coli. Также предложен рекомбинантный штамм Escherichia coli KRX/pMBP-PUUV_N, полученный путем трансформации клеток штамма Escherichia coli KRX указанной плазмидной ДНК pMBP-PUUV_N и продуцирующий гибридный полипептид MBP-PUUV_N, используемый для получения рекомбинантного белка PUUV_N, применяемого для диагностики геморрагической лихорадки с почечным синдромом (ГЛПС). Изобретение обеспечивает синтез рекомбинантного белка МВР-PUUV_N, из которого получают рекомбинантный белок PUUV_N с чистотой препарата не менее 95% для диагностики ГЛПС. 2 н.п. ф-лы, 6 ил., 1 табл., 6 пр.
1. Рекомбинантная плазмидная ДНК pMBP-PUUV_N, обеспечивающая синтез и секрецию слитого рекомбинантного белка МВР-PUUV_N в клетках Е. coli, имеющая размер 7916 п.н. и содержащая следующие элементы генетической конструкции в соответствии с физической и генетической картой:
- ген, кодирующий сигнальную аминокислотную последовательность мальтоза-связывающего белка (МВР), имеющий координаты с 92 по 1192 п.н.;
- в открытой рамке считывания с МВР ген рекомбинантного нуклеопротеина вируса Пуумала (PUUV_N), с нуклеотидной последовательностью SEQ ID NO: 1, имеющий координаты с 1319 по 2614 п.н.;
- нуклеотидную последовательность в рамке считывания гена МВР, кодирующую аминокислотные остатки 6 гистидинов (6-His), имеющую координаты с 1265 по 1282 п.н.;
- нуклеотидную последовательность в рамке считывания гена МВР сайта узнавания и разрезания TEV-протеазой с 1295 по 1315 п.н.;
- промотор и терминатор транскрипции Т7-РНК-полимеразы, имеющие координаты с 4 по 22 п.н. и с 2727 по 2774 соответственно;
- ген β-лактамазы (AmpR), в качестве генетического маркера, определяющего устойчивость к пенициллиновым антибиотикам клеток бактерии Е. сoli, имеющий координаты с 3398 по 3466 п.н.;
- уникальные сайты рестрикции XbaI, NdeI, HindIII, XhoI, имеющие координаты 50, 90, 2622, 2637, соответственно;
- lacI, имеющий координаты с 6447 по 7529 п.н.;
- lacI промотор, имеющий координаты с 7530 по 7607 п.н.;
- ori, имеющий координаты с 4429 по 5017 п.н.;
- fl ori, имеющий координаты с 2811 по 3266 п.н.;
- сайт рестрикции EagI-NotI, имеющий координаты 2629 п.н.;
- lac оператор, имеющий координаты с 23 по 47 п.н.;
- AmpR промотор, имеющий координаты с 3293 по 3397 п.н.
2. Рекомбинантный штамм Escherichia coli KRX/pMBP-PUUV_N, полученный путем трансформации клеток штамма Escherichia coli KRX рекомбинантной плазмидной ДНК pMBP-PUUV_N по п. 1, продуцирующий гибридный полипептид MBP-PUUV_N, используемый для получения рекомбинантного белка PUUV_N, применяемого для диагностики геморрагической лихорадки с почечным синдромом.
| WO 1998014585 А1, 09.04.1998 | |||
| Способ получения серы | 1935 |
|
SU44539A1 |
| N protein [Orthohantavirus puumalaense] | |||
| Способ получения серы | 1935 |
|
SU44539A1 |
| Рекомбинантная плазмида pET21-GST-CD, обеспечивающая синтез и секрецию эктодомена Gc гликопротеина вируса Хантаан, и рекомбинантный штамм Escherichia coli BL21(DE3)pLysE pET21-GST-CD - продуцент белка Gc - эктодомена гликопротеина вируса Хантаан | 2023 |
|
RU2807520C1 |
| СПОСОБ ПОЛУЧЕНИЯ РЕКОМБИНАНТНОГО АНТИГЕНА G2 ХАНТАВИРУСА ДОБРАВА В КЛЕТКАХ E. coli | 2011 |
|
RU2495938C2 |
| СИНГЕР М., БЕРГ П | |||
| Гены и | |||
Авторы
Даты
2025-04-23—Публикация
2023-11-22—Подача