Область техники
Изобретение относится к биотехнологии, в частности, к штаммам микроорганизмов, в частности, к штамму бактерий Escherichia coli - продуценту рекомбинантного неструктурного белка NS1 вируса гриппа А, подходящего для его использования в качестве мишени при разработке новых подходов направленной противовирусной терапии.
Предшествующий уровень техники
Белок NS1 является ключевым антагонистом системы интерферонов и участвует во множественном взаимодействии с клеточными белками, уровень его продукции рассматривается как один из главных факторов, определяющих патогенность вируса гриппа А. Консервативность белка NS1 в ходе эволюции вируса гриппа позволяет использовать этот белок качестве мишени при разработке новых подходов к противовирусной терапии.
Восьмой сегмент вируса гриппа А кодирует как минимум два неструктурных белка NS1 и NS2 (NEP), соотношение продукции которых регулируется сплайсингом. Первоначально с вРНК NS сегмента осуществляется транскрипция непрерывной мРНК, с которой далее запускается продукция NS1. Синтез белка NS1 начинается на ранних этапах заражения, и уровень продукции постоянно увеличивается в течение всего жизненного цикла вируса [1]. В то же время NEP детектируется на более поздних этапах вирусного цикла несмотря на то, что соотношение сплайсированной и несплайсированной форм мРНК гена NS поддерживается на постоянном уровне в течение заражения [2]. Согласно опубликованным данным, предполагается, что баланс уровней белков NS1 и NEP имеет решающее значение в координации вирусного заражения [3]. В настоящее время показано, что оба белка входят в состав вириона вируса гриппа [4].
Белок NS1 вируса гриппа А является многофункциональным неструктурным белком, который посредством взаимодействия с вирусными и клеточными белками участвует в регуляции процессов сплайсинга, экспорта и трансляции вирусных мРНК, обеспечивает противодействие клеточному ответу на инфекцию и выводит из строя целый спектр клеточных механизмов защиты [5]. Белок NS1 относится к факторам вирулентности и межвидовой адаптации вируса, что обусловлено его влиянием на иммунный ответ, а также на эффективность репликации и степень патогенности вируса [6].
Последовательность NS1 является консервативной и варьирует в пределах от 215 до 237 аминокислот, так как встречаются штаммы вируса гриппа А с укороченной С-концевой областью [6]; [7]. Белок состоит из N-концевого РНК-связывающего домена (RBD, RNA-binding domain, 73 а.о.), линкерного домена (LR, linker domain, 10-15 а.о.), эффекторного домена (ED, effector domain, 88-202 а.о.) и небольшого неструктурированного С-концевого хвоста (СТТ, C-terminal tail, 11-33 а.о.) [5]. RBD образован 3 α-спиралями и в форме гомодимеров распознает двунитевую РНК [8]. ED состоит из 3 α-спиралей и 7 β-нитей и осуществляет взаимодействие NS1 с ядерными клеточными белками [9]. Триптофан в положении 187 ED играет ключевую роль в олигомеризации белков NS1 [6]. LD обеспечивает пространственную ориентацию RBD и ED, которая изменяется в течение инфекции и может влиять на переключение активностей NS1 [10].
В клетке NS1 в основном локализуется в ядре, однако на более поздних этапах инфекции белок также обнаруживается в цитоплазме [6]. Предполагается, что внутриклеточное распределение NS1 зависит от доступности сигнальных последовательностей во время его взаимодействия с клеточными белками, а также посттрансляционных модификаций [5]. Высококонсервативная последовательность сигнала ядерной локализации (NLS, nuclear localization signal) в RBD-домене распознается клеточным белком импортином-α и обеспечивает активный транспорт NS1 в ядро [11]; [12]. Для некоторых штаммов также характерно наличие дополнительного NLS и конкурирующего с ним сигнала ядрышковой локализации (NoLS, nucleolar localization sequence) на C-конце белка [12]. В положении 138-147 а.о. располагается последовательность сигнала ядерного экспорта (NES, nuclear export signal) [13].
Подавление иммунного ответа зараженной клетки также происходит в результате множественных взаимодействий белка NS1 с различными сигнальными системами клетки. Двунитевая РНК (днРНК) в цитоплазме является ключевым сигналом для активации противовирусного ответа клетки, в том числе индукции интерферонов (IFN). Белок NS1 напрямую и через связывание с вирусной РНК ингибирует способность внутриклеточных сенсоров, таких как протеинкиназа PKR [14], RIG-1-подобные рецепторы [15]; [16] и 2'-5'-олигоаденилатсинтетаза (OAS, 2'-5'-oligoadenylate synthase) [17], осуществлять распознавание днРНК. Кроме того, NS1 блокирует клеточные белки, регулирующие активацию внутриклеточных сенсоров, в том числе белковый активатор протеинкиназы PACT [18], убиквитинлигазу TRIM25 [19] и антивирусный фактор NF90 [20]. Таким образом, многочисленные взаимодействия NS1 с клеточными белками позволяют вирусу остаться незамеченным в инфицированной клетке, создавая условия для эффективной репликации вируса.
Важным компонентом врожденного ответа является экспрессия интерферонов, которые секретируются инфицированными клетками в ответ на проникновение вируса. NS1 является основным антагонистом системы интерферонов I типа (INF-α/β). Это было установлено благодаря развитию метода обратной генетики, когда удалось получить и охарактеризовать рекомбинантные вирусы, несущие делецию гена NS1 (delNS1) [21] либо его усеченную форму [22]. Такие вирусы проявляли аттенуированные свойства и вызывали более высокие уровни экспрессии интерферонов по сравнению со штаммами ВГА дикого типа в интерферон-компетентных системах. В интерферон-дефицитных живых системах, таких как клеточная культура Vero, развивающиеся куриные эмбрионы и STAT1-нокаутные мыши (delNS1), рекомбинантные и дикие вирусы проявляли одинаковые инфекционные свойства [21]; [22].
Ингибирование индукции интерферонов осуществляется на ко- и пост-транскрипционном уровнях экспрессии. NS1 ингибирует пути активации транскрипционных факторов IRF-3 [23], NF-kB [24] и AP-1 [25], необходимые для инициации транскрипции IFN-мРНК. В белке NS1 вирусов A/H3N2 была идентифицирована H3-гистонподобная последовательность, которая по принципу молекулярной мимикрии взаимодействует с PAF1-комплексом элонгации транскрипции (PAF1C) и вызывает супрессию клеточных генов [26]. Кроме того, было показано, что белок NS1 способен неспецифично связываться с клеточной ДНК и тем самым нарушать процесс сборки клеточной машинерии транскрипции [27].
Как упоминалось выше, NS1 негативно влияет на процессинг и транспорт клеточных мРНК. Белок взаимодействует с 30-кДа субъединицей специфического фактора расщепления и полиаденилирования (CPSF, cleavage and polyadenylation specificity factor) [28]; [29] и поли(А)-связывающим белком PABPII [30], предотвращая таким образом полиаденилирование клеточных мРНК. Данный процесс не нарушает эффективность экспрессии вирусных генов, так как в созревании 3'-концов вирусных мРНК не задействована клеточная машинерия. Кроме того, NS1 напрямую взаимодействует с компонентами сплайсосомы: с U2 и U6 малыми ядерными РНК [31] и с белком-регулятором сплайсинга SF2 [32]. Предполагается, что NS1 нарушает сборку сплайсосомы на клеточной мРНК и регулирует сплайсинг вирусных мРНК [33]; [34]. Блокировка транспорта хозяйских мРНК из ядра происходит за счет формирования комплекса NS1 с белками клеточного экспорта мРНК: NXF-1, RAE-1, p15, Nup98 и E1B-AP5 [35].
Запуск апоптоза зараженной клеткой является одним из ключевых противовирусных механизмов, направленных на ограничение вирусной репликации. Известно, что вирусы используют различные стратегии для задержки апоптоза клеток на ранних этапах инфекции [6]. Согласно опубликованным исследованиям, NS1 активирует фосфатидилинозитол-3-киназный сигнальный путь (PI3K, phosphoinositide 3-kinase), который препятствует преждевременному апоптозу и тем самым способствует прохождению полного цикла вирусной репликации [36]. Ранее было продемонстрировано, что NS1 взаимодействует с регуляторной субъединицей p85β PI3K посредством прямого связывания либо при образовании комплекса NS1/PI3K/Crk (Crkl) через N-концевой SH3-домен белков Crk/Crkl [37]. NS1 также повышает выживаемость клеток с помощью взаимодействия с серин/треонин протеинкиназой Akt и белком p53 [38]; [39]. В противовес антиапоптотическим свойствам NS1 способен связываться с белком теплового шока HSP90 [40] и α-тубулином [41], что, наоборот, может способствовать запуску апоптоза.
Другая роль белка NS1 заключается в усилении трансляции вирусных мРНК. NS1 селективно распознает консервативные последовательности на 5'-UTR, а также взаимодействует с участвующими в трансляции клеточными белками, такими как фактор трансляции eIF4GI [42], поли(А)-связывающий белок PABPI [43] и днРНК-распознающий белок hStaufen, обеспечивающий транспорт мРНК к активным местам трансляции [44].
Известна MDCK клетка-продуцент белков вируса гриппа [45], содержащая лентивирусный плазмидный вектор pLenti6.3/TO/HA(H1N1)-M1-NA, содержащий гены гемагглютинина, нейраминидазы и M1 белка вируса гриппа (H1N1). Описана MDCK клетка-продуцент белков вируса гриппа, содержащая лентивирусный плазмидный вектор pLenti6.3/TO/HA(H3N2)-M1-NA, содержащий клонированный ген гемагглютинина из штамма субтипа H3N2, а также гены нейраминидазы и M1 белка штамма субтипа H1N1. Описана MDCK клетка-продуцент белков вируса гриппа, содержащая лентивирусный плазмидный вектор pLenti6.3/TO/HA(B-)-M1-NA, содержащий ген гемагглютинина вируса гриппа В, а также гены нейраминидазы и Ml белка вируса гриппа Н1N1.
Технической проблемой является необходимость разработки штамма-продуцента рекомбинантного неструктурного белка NS1 вируса гриппа А, а также в расширении ассортимента средств получения неструктурного белка NS1 вируса гриппа А.
Технический результат состоит в получении штамма бактерий Escherichia coli BL-21(DE3)-pET22b(+)-NS1 - продуцента рекомбинантного неструктурного белка NS1 вируса гриппа А.
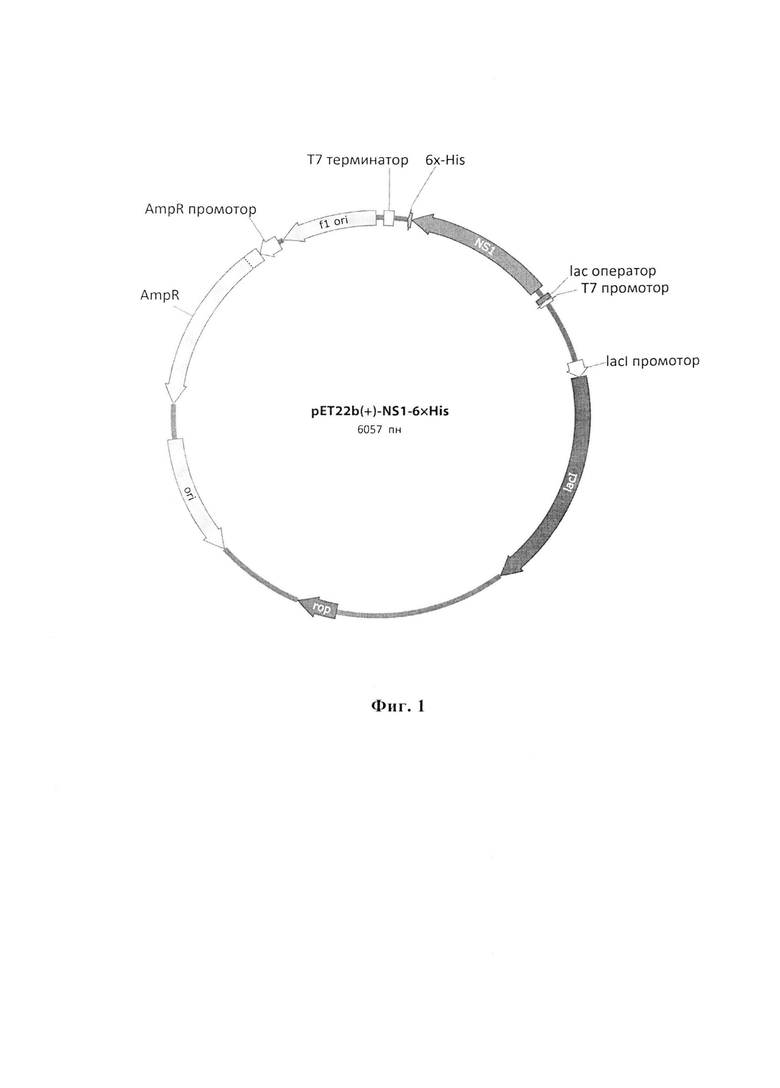
Технический результат достигается тем, что предложена рекомбинантная плазмидная ДНК pET-22b(+)-NS1-6×His для экспрессии в клетках E. coli рекомбинантного неструктурного белка NS1 вируса гриппа А, имеющая карту плазмиды, как показано на Фиг. 1, включающая следующие участки:
- ген β-лактамазы (AmpR), определяющий устойчивость бактериальных клеток к ампициллину;
- участок инициации репликации (ori);
- ROP ген, регулирующий копийность плазмиды;
- фрагмент, содержащий T7 терминатор транскрипции;
- транскрипционный репрессор LacI, регулирующий транскрипцию T7lac-промотора;
- NdeI/XhoI фрагмент, содержащий искусственный ген NS1;
- NdeI/XhoI фрагмент, кодирующий рекомбинантный неструктурный белок NS1 вируса гриппа А, в котором нуклеотидная последовательность соответствует SEQ ID NO:1.
Технический результат также достигается тем, что предложен штамм Escherichia coli BL-21(DE3)-pET22b(+)-NS1 - продуцент рекомбинантного белка с последовательностью NS1, при этом штамм получен путём трансформации клеток штамма E. coli BL-21(DE3) рекомбинантной плазмидной ДНК pET22b(+)-NS1, имеющей карту, показанную на Фиг. 1. Данный штамм был депонирован в Биоресурсный Центр Всероссийской Коллекции Промышленных Микроорганизмов (БРЦ ВКПМ) НИЦ «Курчатовский институт» - ГосНИИгенетика под № В-14117.
Описание фигур
Фиг. 1. Карта рекомбинантной плазмидной ДНК pET-22b(+)-NS1-6×His.
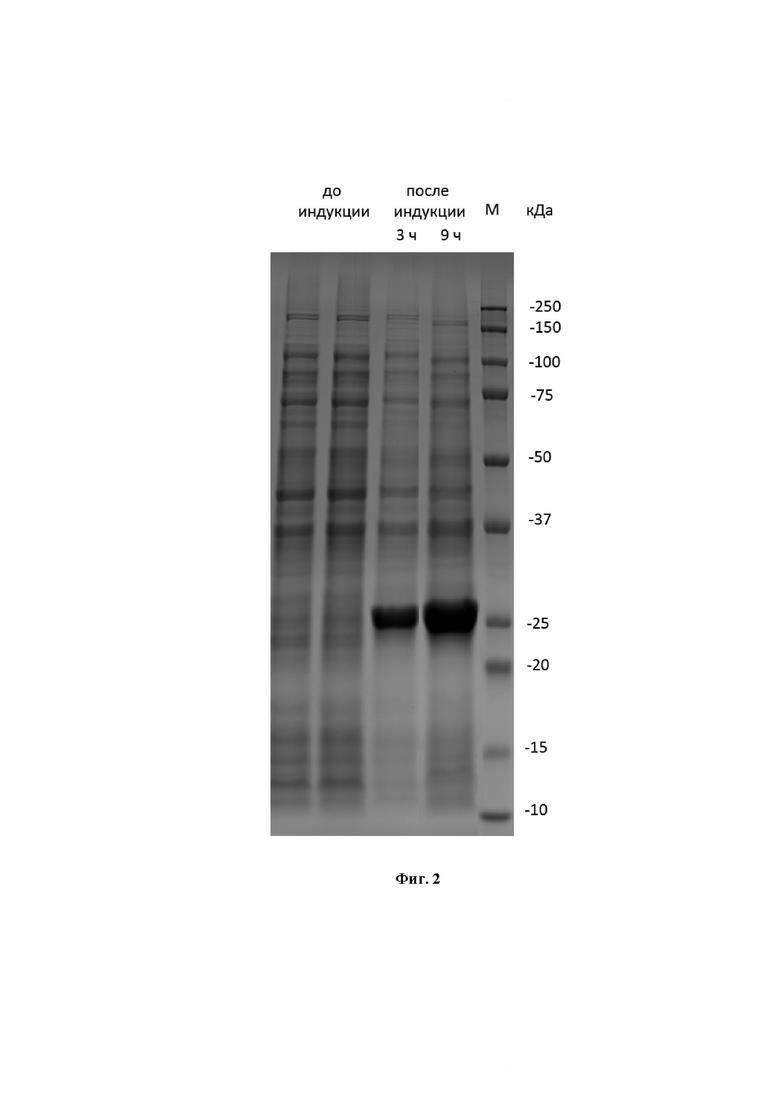
Фиг. 2. Электрофореграмма фракций лизатов штамма-продуцента NS1 (E. coli BL-21). Продукция рекомбинантного белка NS1-6×His. Клеточный лизат до добавления индуктора, клеточный лизат через 3 ч и через 9 ч после добавления индуктора.
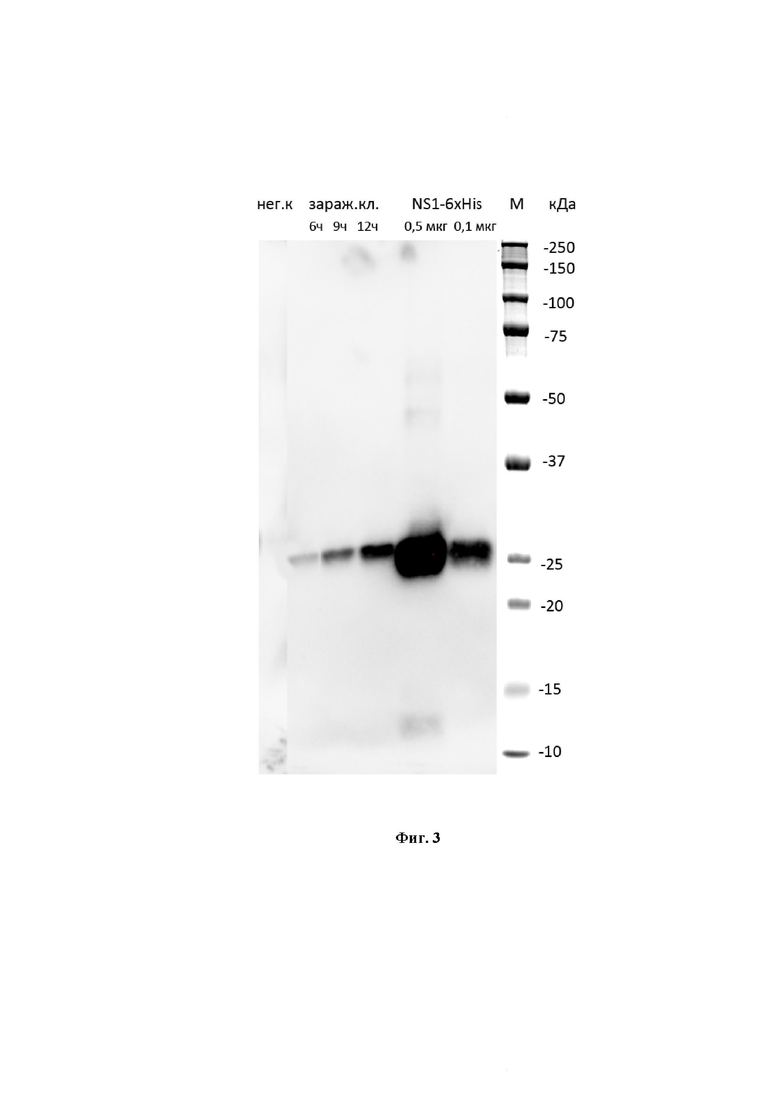
Фиг. 3. Определение белка NS1 в зараженных клетках A549 и NS1-6×His методом иммуноблотинга. Дорожки: №1 - незараженные клетки А549; № 2, 3, 4 - клетки А549, зараженные лабораторным штаммом вируса гриппа A/Puerto Rico/8/1934 H1N1 в дозе, соответствующей 1TИД50/клетку через 3, 6, 9 часов после заражения; №5 - 0,5 мкг рекомбинантного белка NS1-6×His; №6 - 0,1 мкг рекомбинантного белка NS1-6×His.
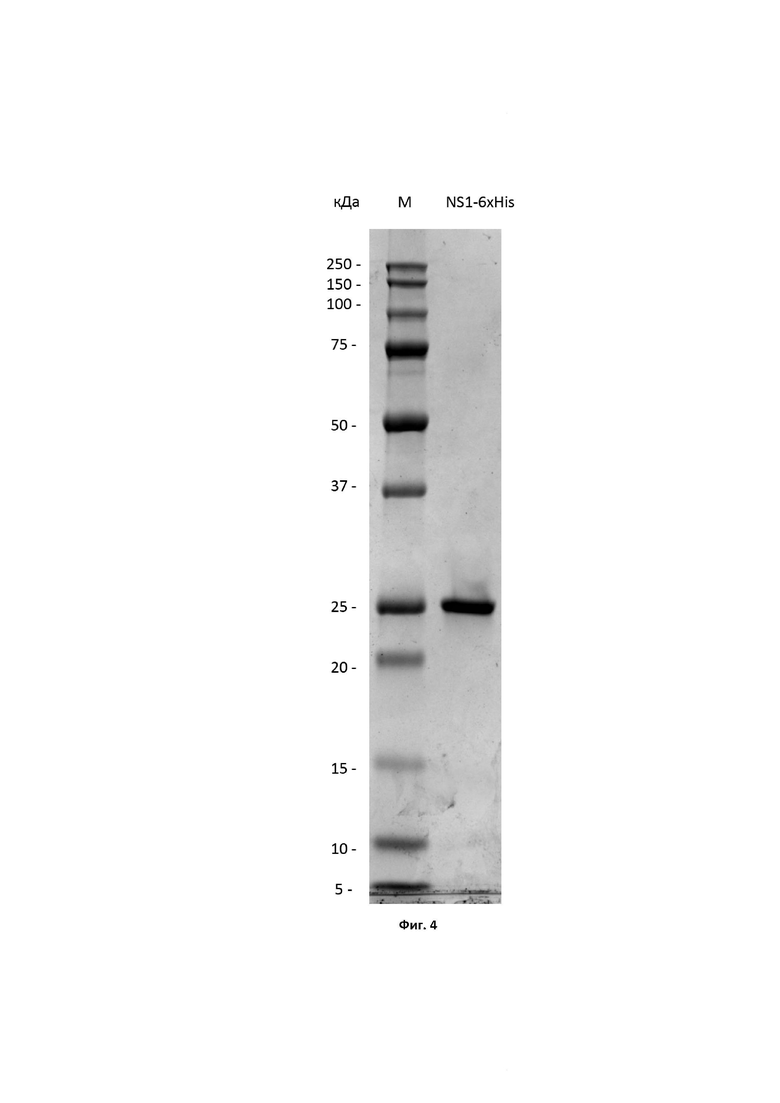
Фиг. 4. Электрофореграмма очищенного рекомбинантного белка NS1. Дорожки: №1 - маркер молекулярной массы белков Precision Plus Protein Unstained, 10-250 кДа (“Bio-Rad”, США), №2 - целевой белок NS1-6×His.
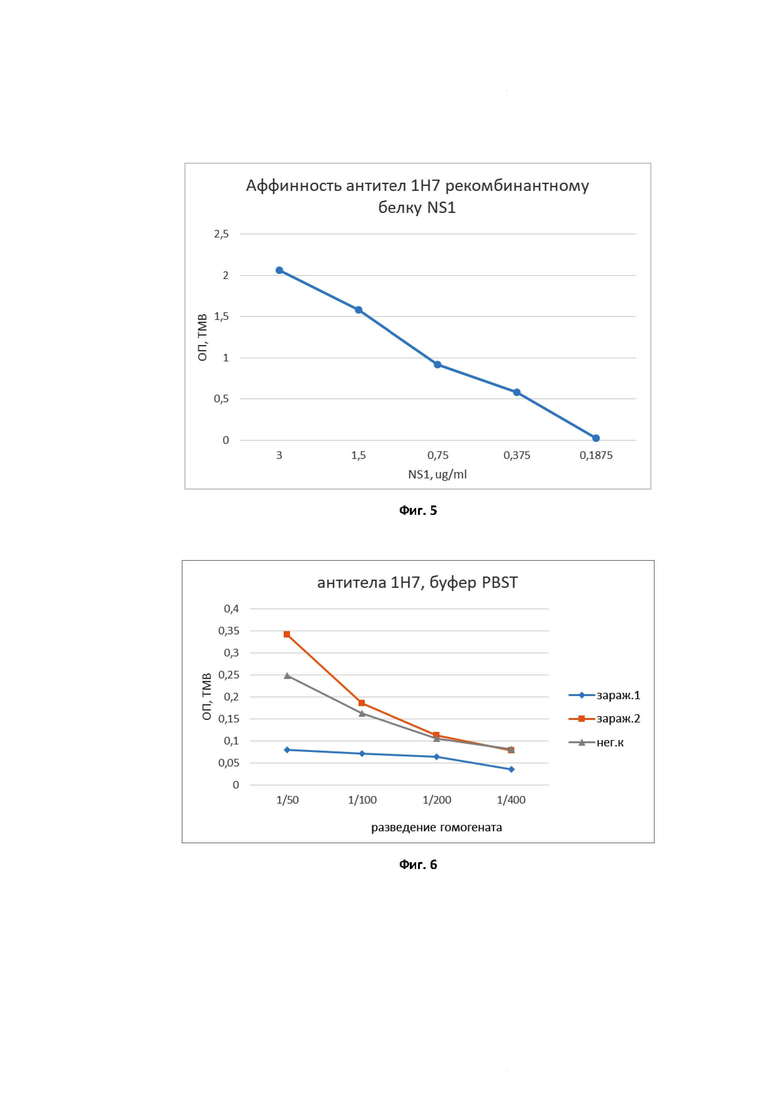
Фиг. 5. Аффинность антител 1H7 к рекомбинантному белку NS1.
Фиг. 6. Определение белка NS1 в легких зараженных мышей методом иммуноферментного анализа по типу сэндвич-метода.
Описание изобретения
Предложена рекомбинантная плазмидная ДНК pET-22b(+)-NS1-6×His для экспрессии в клетках E. coli рекомбинантного неструктурного белка NS1 вируса гриппа А, имеющая карту плазмиды, как показано на Фиг. 1, включающая следующие участки:
- ген β-лактамазы (AmpR), определяющий устойчивость бактериальных клеток к ампициллину;
- участок инициации репликации (ori);
- ROP ген, регулирующий копийность плазмиды;
- фрагмент, содержащий T7 терминатор транскрипции;
- транскрипционный репрессор LacI, регулирующий транскрипцию T7lac-промотора;
- NdeI/XhoI фрагмент, содержащий искусственный ген NS1;
- NdeI/XhoI фрагмент, кодирующий рекомбинантный белок NS1, в котором нуклеотидная последовательность соответствует SEQ ID NO:1.
Кроме того, согласно изобретению, предложен штамм Escherichia coli BL-21(DE3)-pET22b(+)-NS1 - продуцент рекомбинантного белка с последовательностью NS1, при этом штамм получен путём трансформации клеток штамма E. coli BL-21(DE3) рекомбинантной плазмидной ДНК pET-22b(+)-NS1-6×His, имеющей карту, показанную на Фиг. 1.
Техническая проблема заключается в необходимости разработки эффективного способа получения штамма-продуцента рекомбинантного неструктурного белка NS1 вируса гриппа А с применением рекомбинантной плазмидной ДНК pET-22b(+)-NS1-6×His и доступных клеток-продуцентов, например, E. coli.
Технический результат состоит в получении штамма-продуцента Escherichia coli BL-21(DE3)-pET22b(+)-NS1, который является продуцентом рекомбинантного неструктурного белка NS1 вируса гриппа А.
Технический результат достигается за счёт того, что путём трансформации клеток Escherichia coli рекомбинантной плазмидной ДНК pET-22b(+)-NS1-6×His получен штамм E. coli BL-21(DE3)-pET22b(+)-NS1, который является продуцентом рекомбинантного неструктурного белка NS1 вируса гриппа А. Данный штамм был депонирован в Биоресурсный Центр Всероссийской Коллекции Промышленных Микроорганизмов (БРЦ ВКПМ) НИЦ «Курчатовский институт» - ГосНИИгенетика под № В-14117.
Также технический результат достигается за счёт того, что для трансформации клеток E. coli с целью получения рекомбинантного неструктурного белка NS1 вируса гриппа А синтезируют и кодируют в рекомбинантную плазмидную ДНК pET-22b(+) оптимизированную in silico последовательность SEQ ID NO:1, кодирующую рекомбинантный неструктурный белок NS1 вируса гриппа А с последовательностью SEQ ID NO:2, причём рекомбинантная плазмидная ДНК содержит последовательность, кодирующую полигистидиновую метку на C-конце синтезируемого белка. Далее получают ДНК-конструкцию pET-22b(+)-NS1-6×His, с помощью которой трансформируют культуру клеток E. coli с последующим получением штамма-продуцента E. coli BL-21(DE3)-pET22b(+)-NS1. Получаемый рекомбинантный неструктурный белок NS1 вируса гриппа А обладает стабильностью при хранении и может быть использован в качестве мишени при разработке новых подходов направленной противовирусной терапии.
Далее способ получения рекомбинантной плазмидной ДНК pET-22b(+)-NS1-6×His и рекомбинантного неструктурного белка NS1 вируса гриппа А будет описан в виде примеров, которые предназначены для иллюстрации изобретения и никоим образом не ограничивают его.
Получение рекомбинантной плазмидной ДНК pET22b(+)-NS1-6×His
Штамм Escherichia coli BL21[DE3]pet22b(+)-NS1 - продуцент рекомбинантного белка с последовательностью NS1 вируса гриппа A/Puerto Rico/8/1934 (H1N1), при этом штамм получен путём электропорации клеток штамма E. coli BL-21(DE3) рекомбинантной плазмидной ДНК pET22b(+)-NS1-6×His, имеющей карту, показанную на Фиг. 1.
Конечная аминокислотная последовательность рекомбинантного белка, кодируемого рекомбинантной плазмидной ДНК pET22b(+)-NS1-6×His, представлена в SEQ ID NO:2.
Фрагмент ДНК, кодирующий ген NS вируса гриппа А, был синтезирован путем полимеразной цепной реакции. Для этого был использован штамм A/Puerto Rico/8/1934 (H1N1), полученный из коллекции вирусов ФГБУ «НИИ гриппа им. А.А. Смородинцева» Минздрава России. Последовательности праймеров, использованные для амплификации целевого фрагмента, содержали сайты рестрикции на 5'-концах: сайт рестрикции Nde I в прямом праймере (5'-AACCTTCATATggATCCAAACACTgTg-3' - SEQ ID NO:3) и сайт рестрикции Xho I в обратном праймере (5'-TTAACTCgAgAACTTCTgACCTAATTgTTC-3' - SEQ ID NO:4). ПЦР была проведена с использованием термоциклера Bio-Rad C1000, высокоточной Q5 ДНК-полимеразы (NEB, #M0491S) и 5× реакционного буфера Q5 (NEB, #B9027S) в соответствии с рекомендациями производителя. Далее было проведено электрофоретическое разделение ПЦР продукта в 1% агарозе в 1-кратном буфере ТВЕ при комнатной температуре с последующей экстракцией ампликона из геля с помощью набора Cleanup Standard («Евроген», BC022).
Для проведения реакции лигирования использовались следующие ферменты: рестриктазы Xho I Fast Digest (Thermo Fisher Scientific, #FD0695) и Nde I Fast Digest (Thermo Fisher Scientific, #FD0585) и ДНК-лигаза Т4 (NEB, #M0202L). Электропорация бактериальной культуры DH5a была выполнена согласно протоколу производителя (Bio-Rad, #1652100). Далее была проведена селекция бактериальных клонов в присутствии антибиотика (ампициллин, 100 мкг/мл). Выделение плазмидной ДНК было проведено с помощью наборов Qiagen (#12943) и Евроген (#BC021) с последующим подтверждением внесенных мутаций секвенированием по Сэнгеру. Процедуру секвенирования проводили с использованием Т7-праймеров в компании «Евроген» (Москва).
Реагенты:
1. Ферменты:
Q5® High-Fidelity DNA Polymerase (NEB, M0491S);
5× Q5 Reaction Buffer (NEB, B9027S)
Xho I Fast Digest (Thermo Fisher Scientific, FD0695)
Nde I Fast Digest (Thermo Fisher Scientific, FD0585)
FastDigest Buffer (10×, Thermo Fisher Scientific, B64)
T4 DNA Ligase (NEB, M0202L)
T4 DNA Ligase Buffer (10×, NEB, B0202S)
2. Плазмиды:
pET-22b+
pHW2006, кодирующая генный сегмент NS вируса гриппа A/Puerto Rico/8/1934(H1N1).
3. Праймеры:
cl-NS1-Nde1-F AACCTTCATATggATCCAAACACTgTg (SEQ ID NO:3)
cl-NS1-Xho1-R TTAACTCgAgAACTTCTgACCTAATTgTTC (SEQ ID NO:4)
4. Бактериальные культуры:
DH5a, BL-21(DE3)
5. Наборы:
Набор для выделения и очистки плазмидной ДНК из культуры E. coli “Plasmid Miniprep” (Евроген, BC021);
Наборы для очистки ДНК из агарозного геля и реакционных смесей “Cleanup Standard” (Евроген, BC022);
Программа ПЦР:
98°C - 30 секунд
98°C - 10 секунд
53°C - 20 секунд
72°C - 20 секунд
25 циклов
72°C - 1 минута
Условия культивирования рекомбинантного неструктурного белка NS1 вируса гриппа А в биореакторе
Исходную посевную культуру клеток E. coli BL-21(DE3), содержащих pET-22b(+)-NS1-6×His инкубировали в течение 8 ч при 37 °C. Для получения инокулята для культивирования в биореакторе 200 мкл посевной культуры переносили в 250 мл-колбы Эрленмейера, содержащие 50 мл среды Лурия-Бертани (LB). Состав среды: 5 г/л дрожжевого экстракта, 10 г/л триптона, 10 г/л NaCl и 100 мг/л ампициллина.
Оптимальные условия получения рекомбинантного белка были получены при культивировании бактериальных клеток в 10-литровом ферментере (BioFlo / CelliGen 115, New Brunswick). Ночная посевная культура была использована для инокуляции 8 л культуральной среды (pH 7,4), содержащей (г/л): гидролизат казеина - 15; дрожжевой экстракт - 7,5; NaCl - 7; глюкоза - 5; NH4Cl - 1; MgSO4 - 0,24; KН2PО4 ×1Н2О - 3; Na2HPO4×2H2O - 14,3; и ампициллин - 0,1. Контроль биопроцесса вели по следующим параметрам: концентрация растворенного кислорода в культуральной среде и pH. Концентрация растворенного кислорода поддерживалась при варьировании скорости потока воздуха и скорости вращения перемешивающего устройства. pH среды постоянно поддерживался в диапазоне значений от 6,9 до 7,0 добавлением 1 M раствора H3PO4 или 2М раствора NaOH. Время добавления в среду дополнительных источников углерода определяли по увеличению концентрации растворенного кислорода и снижению pH в культуральной среде. Затем температуру понижали до 32°C и добавляли IPTG до концентрации 1 мМ, а затем добавляли к культуральной среде дополнительные источники углерода (гидролизат казеина - 1,5 г/л, дрожжевой экстракт - 0,75 г/л и глицерин 6 г/л) и продолжали вести культивирование. После добавления индуктора процесс культивирования вели 3 часа. Общая продолжительность культивирования в биореакторе составила 8 часов.
Очистка и рефолдинг рекомбинантного неструктурного белка NS1 вируса гриппа А
Хроматографическую очистку рекомбинантного неструктурного белка NS1 вируса гриппа А проводили с помощью металл-аффинной хроматографии с последующей заменой буфера путём гель-фильтрации на хроматографической системе AKTA Start производства GE Healthcare. Клетки осаждали центрифугированием, далее, влажный осадок ресуспендировали в денатурирующем буфере (30 мМ фосфат натрия, pH 7.5, 750 мМ хлорид натрия, 20 мМ имидазол, 1 мМ PMSF), в соотношении 10 мл буфера на 1 г клеток, и обрабатывали суспензию ультразвуком на ледяной бане (ультразвуковой гомогенизатор MSE). Проводили 10 циклов: на 1 минуту включали, на 1 минуту выключали. Далее, полученный лизат осветляли центрифугированием в течение 1 часа при +4°C с ускорением 13000 g (центрифуга Eppendorf 5810R).
Хроматографическую колонку HisTrap FF Crude объёмом 1 мл (GE Healthcare, США) уравновешивали 5 мл стартового буфера (30 мМ фосфат натрия, 750 мМ хлорид натрия, 20 мМ имидазол, pH 7,5, 1 мМ PMSF) на скорости потока 1 мл/мин. Далее, осветлённый лизат вносили в колонку на той же скорости потока, после чего колонку отмывали 20 мл стартового буфера на скорости потока 1 мл/мин. Целевой белок элюировали 10 мл элюирующего буфера (30 мМ фосфат натрия, 750 мМ хлорид натрия, 500 мМ имидазол, pH 7,5) на скорости потока 1 мл/мин, в ходе элюции собирали фракции по 0,5 мл. Фракции, содержащие целевой белок, идентифицировали путём денатурирующего электрофореза в ПААГ, объединяли и пропускали через шприцевой фильтр Sartorius (диаметр 33 мм, мембрана PES, размер пор 0,22 мкм). Рекомбинантный неструктурный белок NS1 вируса гриппа А дополнительно очищали гель-фильтрацией с использованием хроматографа AKTA Pure (GE Healthcare, США). Хроматографическую колонку XK 16/60, заполненную 120 мл сорбента Sephadex S-200 HR (диапазон фракционирования глобулярных белков 5-250 кДа), промывали последовательно 120 мл дистиллированной воды и 240 мл 2× PBS. Затем рекомбинантный белок вводили в колонку с помощью 5-миллилитровой петли и элюировали 180 мл 2× PBS. В ходе элюции собирали фракции по 2,5 мл. Присутствие белка в элюате контролировали по поглощению на длине волны 280 нм с использованием проточного УФ-детектора с длиной оптического пути 2 мм. Фракции анализировали путём денатурирующего электрофореза в ПААГ. Фракции, содержащие чистый целевой белок без примесей, объединяли. Калибровку колонки проводили с использованием смеси стандартных белков (GE Gel Filtration Calibration Kit) в соответствии с инструкцией производителя.
Анализ полученного рекомбинантного неструктурного белка NS1 вируса гриппа А
ПААГ-электрофорез
Анализ чистоты полученного рекомбинантного неструктурного белка NS1 вируса гриппа А проводили методом ПААГ-электрофореза по методу Лэммли в восстанавливающих условиях [46]. Для этого использовали предзалитые гели TGX anyKD, источник постоянного тока PowerPac Basic и камеру для ПААГ-электрофореза Mini Protean Tetra производства Bio-Rad. Гель окрашивали раствором красителя Кумасси как описано в [47], после чего отмывали и визуализировали при помощи системы гель-документации ChemiDoc MP и анализировали при помощи программного обеспечения Image Lab Software производства Bio-Rad. Теоретическая масса белка составляет 25 кДа. На Фиг. 2 представлен препарат до проведения хроматографической очистки, на Фиг. 4 представлен очищенный рекомбинантный белок NS1 вируса гриппа А.
MALDI-TOF
Аминокислотная последовательность рекомбинантного неструктурного белка NS1 вируса гриппа А SEQ ID NO:2 была подтверждена с помощью MALDI-TOF. Фрагменты геля вырезали и дважды отмывали от красителя, используя раствор, содержащий 30 мМ NH4HCO3 и 40% ацетонитрила. После этого кусок геля обезвоживали 100% ацетонитрилом, сушили на воздухе и подвергали ферментативному расщеплению трипсином или химотрипсином. К гелю добавляли трипсин (20 мкг/мл в 50 мМ NH4HCO3) и проводили инкубацию в течение 2 ч в при 60°С. Инкубацию с химотрипсином (20 мкг/мл в 100 мМ Трис-HCl, 1 мМ CaCl2, pH 8,0) проводили в течение 18 ч при 25°C. В обоих случаях реакцию останавливали 1% ТФУ, 10% ацетонитрила. Пептиды после ферментативного расщепления смешивали с матрицей HCCA, наносили на металлическую мишень и масс-спектры записывали в отражательном режиме регистрации положительных ионов с использованием масс-спектрометра MALDI-TOF UltrafleXtreme (Bruker). Спектры обрабатывали в программе flexAnalysis (Bruker). Идентификацию белков проводили в BioTools (Bruker) с использованием MASCOT (http://www.matrixscience.com/). Следует отметить, что аминокислотная последовательность рекомбинантного белка SEQ ID NO:2 была добавлена в локальную базу данных, и в процессе идентификации одновременно использовались две базы данных (локальная и SwissProt, https://www.uniprot.org/). Допуск по массе был установлен как 20 частей на миллион, окисление метионина обозначено как изменяемая модификация. Идентификация считалась достоверной в случае, если Score спектров превышал пороговое значение (p <0,05).
ИФА
На дно планшета сорбировали антитела 1H7 (любезно предоставленные лабораторией биотехнологии диагностических препаратов ФГБУ «НИИ гриппа им. А.А. Смородинцева» Минздрава России). После блокировки 5%-ным молоком в лунки планшета добавляли исследуемые образцы легочной суспензии, разведенные в 50, 100, 200, и 400 раз в буфере PBST (PBS, содержащий 0.1% Tween-20), а также последовательные двукратные разведения полученного рекомбинантного белка NS1-6×His. Для выявления белка NS1 была использована высокотитражная кроличья антисыворотка к белку NS1 (полученная в рамках предыдущих работ) и меченный пероксидазой хрена конъюгат Goat Anti-Rabbit IgG (H+L)-HRP Conjugate (Bio-Rad, #1706515) (Фиг. 5).
Иммуноблотинг
Cуточный монослой клеток А549 в 6-луночных планшетах заражали вирусом A/Puerto Rico/8/1934 (H1N1), в дозе 1.0 ТИД50/клетку, в трех независимых повторах для каждого вируса. Через 3, 6 и 9 часов после заражения было проведено аккуратное снятие клеток с помощью скребка и их последующее растворение в PBS. Измерение концентрации тотального белка было выполнено с использованием метода Лоури c помощью набора «DC Protein Assay Kit I» (Bio-Rad, #5000111). Образцы были смешаны с буфером Лэммли, содержащим бета-меркаптоэтанол, и проведена денатурация белков при 95°С в течение 5 минут в твердотельном термостате «Гном» («ДНК-Технология», Россия). В лунки ПААГ «Any kD Mini-PROTEAN TGX Stain-Free precast Protein Gel» (Bio-Rad, #4568121) были нанесены 0,5 мкг белка каждого образца зараженных клеток и 0,5 / 0,1 мкг рекомбинантного белка NS1-6×His. Разделение белков проводилось в электрофоретической ячейке Mini-PROTEAN Tetra Vertical Electrophoresis Cell (Bio-Rad) при следующих параметрах: 25 мА на каждый гель, 220 В. Клеточные белки разделяли методом электрофореза в ПААГ, после чего переносили на нитроцеллюлозную мембрану и окрашивали моноклональными антителами 1Н7 к NS1 [48]. Для последующей визуализации белков был использован конъюгат меченных пероксидазой хрена козьих антител к мышиным IgG (Goat Anti-Mouse IgG (H+L)-HRP Conjugate, Bio-Rad #1721011). Мембраны были проявлены набором Clarity Western ECL Substrate (Bio-Rad). Фотографию мембран получали с использованием системы визуализации ChemiDoc MP (Bio-Rad) (Фиг. 3).
Заражение лабораторных животных
Самки мышей линий BALB/c (массой 17-20 грамм) в возрасте 6-8 недель были получены из питомника «Столбовая» ФГБУН «Научный центр биомедицинских технологий» ФМБА России. Животные были заражены лабораторным штаммом вируса гриппа A/Puerto Rico/8/1934 (H1N1) в дозе 10 МИД50 (рассчитанной исходя из значения МИД50 для данной партии мышей). На второй день после заражения животных подвергали эвтаназии и извлекали легкие. Гомогенизация легких животных была выполнена с помощью прибора Tissue Lyser II (Qiagen) в стерильном растворе DPBS (Биолот). Осветленную центрифугированием 10%-ную суспензию (3000 об/мин, 10 мин) использовали для определения белка NS1 методом ИФА (Фиг. 6).
Список литературы
1. Robb, Nicole C., David Jackson, Frank T. Vreede, and Ervin Fodor. 2010. “Splicing of Influenza A Virus NS1 MRNA Is Independent of the Viral NS1 Protein.” Journal of General Virology 91(9): 2331-40.
2. Huang, Xiaofeng et al. 2017. “An NS-Segment Exonic Splicing Enhancer Regulates Influenza A Virus Replication in Mammalian Cells.” Nature Communications 8: 1-15.
3. Chua, Mark A. et al. 2013. “Influenza A Virus Utilizes Suboptimal Splicing to Coordinate the Timing of Infection.” Cell Reports 3(1): 23-29. http://dx.doi.org/10.1016/j.celrep.2012.12.010.
4. Eisfeld, Amie J., Gabriele Neumann, and Yoshihiro Kawaoka. 2015. “At the Centre: Influenza A Virus Ribonucleoproteins.” Nature Reviews Microbiology 13(1): 28-41.
5. Klemm, Carolin, Yvonne Boergeling, Stephan Ludwig, and Christina Ehrhardt. 2018. “Immunomodulatory Nonstructural Proteins of Influenza A Viruses.” Trends in Microbiology 26(7): 624-36.
6. Hale, B. G., R. E. Randall, J.  , and D. Jackson. 2008. The Multifunctional NS1 Protein of Influenza A Viruses. Society for General Microbiology.
, and D. Jackson. 2008. The Multifunctional NS1 Protein of Influenza A Viruses. Society for General Microbiology.
7. Васин, А.В. et al. 2017. “Эволюционная Динамика Структурных и Функциональных Доменов Белка Ns1 Вирусов Гриппа а Человека.” Вопросы вирусологии 62(15): 246-58.
8. Hale, Benjamin G. 2014. “Conformational Plasticity of the Influenza A Virus NS1 Protein.” Journal of General Virology 95(10): 2099-2105. https://www.microbiologyresearch.org/content/journal/jgv/10.1099/vir.0.066282-0 (October 25, 2020).
9. Bornholdt, Zachary A., and B. V.Venkataram Prasad. 2006. “X-Ray Structure of Influenza Virus NS1 Effector Domain.” Nature Structural and Molecular Biology 13(6): 559-60. https://www.nature.com/articles/nsmb1099 (October 26, 2020).
10. Carrillo, B. et al. 2014. “The Influenza A Virus Protein NS1 Displays Structural Polymorphism.” Journal of Virology 88(8): 4113-22. http://jvi.asm.org/ (October 25, 2020).
11. Greenspan, D, P Palese, and M Krystal. 1988. “Two Nuclear Location Signals in the Influenza Virus NS1 Nonstructural Protein.” Journal of Virology 62(8): 3020-26.
12.  , Krister et al. 2007. “Nuclear and Nucleolar Targeting of Influenza A Virus NS1 Protein: Striking Differences between Different Virus Subtypes.” Journal of Virology 81(11): 5995-6006. http://jvi.asm.org/ (October 25, 2020).
, Krister et al. 2007. “Nuclear and Nucleolar Targeting of Influenza A Virus NS1 Protein: Striking Differences between Different Virus Subtypes.” Journal of Virology 81(11): 5995-6006. http://jvi.asm.org/ (October 25, 2020).
13. Li, Yongzhong, Yoshihiko Yamakita, and Robert M. Krug. 1998. “Regulation of a Nuclear Export Signal by an Adjacent Inhibitory Sequence: The Effector Domain of the Influenza Virus NS1 Protein.” Proceedings of the National Academy of Sciences of the United States of America 95(9): 4864-69. www.pnas.org. (October 25, 2020).
14. Min, Ji Young, Shoudong Li, Ganes C. Sen, and Robert M. Krug. 2007. “A Site on the Influenza A Virus NS1 Protein Mediates Both Inhibition of PKR Activation and Temporal Regulation of Viral RNA Synthesis.” Virology 363(1): 236-43.
15. Guo, Zhu et al. 2007. “NS1 Protein of Influenza A Virus Inhibits the Function of Intracytoplasmic Pathogen Sensor, RIG-I.” American Journal of Respiratory Cell and Molecular Biology 36(3): 263-69. http://www.atsjournals.org/doi/abs/10.1165/rcmb.2006-0283RC (October 26, 2020).
16. Mibayashi, Masaki et al. 2007. “Inhibition of Retinoic Acid-Inducible Gene I-Mediated Induction of Beta Interferon by the NS1 Protein of Influenza A Virus.” Journal of Virology 81(2): 514-24. http://jvi.asm.org/ (October 26, 2020).
17. Min, Ji Young, and Robert M. Krug. 2006. “The Primary Function of RNA Binding by the Influenza A Virus NS1 Protein in Infected Cells: Inhibiting the 2′-5′ Oligo (A) Synthetase/RNase L Pathway.” Proceedings of the National Academy of Sciences of the United States of America 103(18): 7100-7105. www.pnas.orgcgidoi10.1073pnas.0602184103 (October 26, 2020).
18. Tawaratsumida, K. et al. 2014. “Quantitative Proteomic Analysis of the Influenza A Virus Nonstructural Proteins NS1 and NS2 during Natural Cell Infection Identifies PACT as an NS1 Target Protein and Antiviral Host Factor.” Journal of Virology 88(16): 9038-48. http://dx.doi.org/10.1128 (October 26, 2020).
19. Rajsbaum, Ricardo et al. 2012. “Species-Specific Inhibition of RIG-I Ubiquitination and IFN Induction by the Influenza A Virus NS1 Protein” ed. Andrew Pekosz. PLoS Pathogens 8(11): e1003059. https://dx.plos.org/10.1371/journal.ppat.1003059 (October 26, 2020).
20. Li, Ting et al. 2016. “NF90 Is a Novel Influenza A Virus NS1-interacting Protein That Antagonizes the Inhibitory Role of NS1 on PKR Phosphorylation.” FEBS Letters 590(16): 2797-2810. https://febs.onlinelibrary.wiley.com/doi/full/10.1002/1873-3468.12311%4010.1002/%28ISSN%291873-3468.FEMS (October 26, 2020).
21.  , Adolfo et al. 1998. “Influenza A Virus Lacking the NS1 Gene Replicates in Interferon-Deficient Systems.” 330: 324-30.
, Adolfo et al. 1998. “Influenza A Virus Lacking the NS1 Gene Replicates in Interferon-Deficient Systems.” 330: 324-30.
22. Egorov, Andrej et al. 1998. “Transfectant Influenza A Viruses with Long Deletions in the NS1 Protein Grow Efficiently in Vero Cells.” 72(8): 6437-41.
23. Talon, Julie et al. 2000. “Activation of Interferon Regulatory Factor 3 Is Inhibited by the Influenza A Virus NS1 Protein.” Journal of Virology 74(17): 7989-96. http://jvi.asm.org/ (October 28, 2020).
24. Wang, Xiuyan et al. 2000. “Influenza A Virus NS1 Protein Prevents Activation of NF-KB and Induction of Alpha/Beta Interferon.” Journal of Virology 74(24): 11566-73. http://jvi.asm.org/ (October 28, 2020).
25. Ludwig, Stephan et al. 2002. “The Influenza A Virus NS1 Protein Inhibits Activation of Jun N-Terminal Kinase and AP-1 Transcription Factors.” Journal of Virology 76(21): 11166-71. http://jvi.asm.org/ (October 28, 2020).
26. Marazzi, Ivan et al. 2012. “Suppression of the Antiviral Response by an Influenza Histone Mimic.” Nature 483(7390): 428-33. https://www.nature.com/articles/nature10892 (October 28, 2020).
27. Anastasina, Maria et al. 2016. “Influenza Virus NS1 Protein Binds Cellular DNA to Block Transcription of Antiviral Genes.” Biochimica et Biophysica Acta - Gene Regulatory Mechanisms 1859(11): 1440-48.
28. Nemeroff, Martin E. et al. 1998. “Influenza Virus NS1 Protein Interacts with the Cellular 30 KDa Subunit of CPSF and Inhibits 3′ End Formation of Cellular Pre-MRNAs.” Molecular Cell 1(7): 991-1000.
29. Noah, Diana L., Karen Y. Twu, and Robert M. Krug. 2003. “Cellular Antiviral Responses against Influenza A Virus Are Countered at the Posttranscriptional Level by the Viral NS1A Protein via Its Binding to a Cellular Protein Required for the 3′ End Processing of Cellular Pre-MRNAS.” Virology 307(2): 386-95.
30. Chen, Zhongying, Yongzhong Li, and Robert M. Krug. 1999. “Influenza A Virus NS1 Protein Targets Poly(A)-Binding Protein II of the Cellular 3'-End Processing Machinery.” EMBO Journal 18(8): 2273-83. https://www.embopress.org/doi/full/10.1093/emboj/18.8.2273 (November 2, 2020).
31. Qiu, Yun M, Martin Nemeroff, and Robert M. Krug. 1995. “The Influenza Virus NS1 Protein Binds to a Specific Region in Human U6 SnRNA and Inhibits U6-U2 and U6-U4 SnRNA Interactions during Splicing.” RNA (New York, N.Y.) 1(3): 304-16.
32. Huang, Xiaofeng et al. 2017. “An NS-Segment Exonic Splicing Enhancer Regulates Influenza A Virus Replication in Mammalian Cells.” Nature Communications 8: 1-15.
33. Robb, Nicole C., and Ervin Fodor. 2012. “The Accumulation of Influenza A Virus Segment 7 Spliced MRNAs Is Regulated by the NS1 Protein.” Journal of General Virology 93(1): 113-18. https://www.microbiologyresearch.org/content/journal/jgv/10.1099/vir.0.035485-0 (October 26, 2020).
34. Tsai, Pei-Ling et al. 2013. “Cellular RNA Binding Proteins NS1-BP and HnRNP K Regulate Influenza A Virus RNA Splicing” ed. Andrew Pekosz. PLoS Pathogens 9(6): e1003460. https://dx.plos.org/10.1371/journal.ppat.1003460 (October 26, 2020).
35. Satterly, Neal et al. 2007. “Influenza Virus Targets the MRNA Export Machinery and the Nuclear Pore Complex.” Proceedings of the National Academy of Sciences of the United States of America 104(6): 1853-58. www.pnas.org/cgi/content/full/ (October 30, 2020).
36. Ehrhardt, Christina et al. 2007. “Influenza A Virus NS1 Protein Activates the PI3K/Akt Pathway To Mediate Antiapoptotic Signaling Responses.” Journal of Virology 81(7): 3058-67. http://jvi.asm.org/ (November 2, 2020).
37. Heikkinen, Leena S. et al. 2008. “Avian and 1918 Spanish Influenza A Virus NS1 Proteins Bind to Crk/CrkL Src Homology 3 Domains to Activate Host Cell Signaling.” Journal of Biological Chemistry 283(9): 5719-27. http://www.jbc.org/ (November 2, 2020).
38. Wang, Xiaodu et al. 2010. “The Non-Structural (NS1) Protein of Influenza A Virus Associates with P53 and Inhibits P53-Mediated Transcriptional Activity and Apoptosis.” Biochemical and Biophysical Research Communications 395(1): 141-45.
39. Zhirnov, Oleg P., and Hans Dieter Klenk. 2007. “Control of Apoptosis in Influenza Virus-Infected Cells by up-Regulation of Akt and P53 Signaling.” Apoptosis 12(8): 1419-32. https://link.springer.com/article/10.1007/s10495-007-0071-y (November 2, 2020).
40. Zhang, Chuanfu et al. 2011. “The NS1 Protein of Influenza a Virus Interacts with Heat Shock Protein Hsp90 in Human Alveolar Basal Epithelial Cells: Implication for Virus-Induced Apoptosis.” Virology Journal 8(1): 1-9. https://link.springer.com/articles/10.1186/1743-422X-8-181 (November 2, 2020).
41. Han, Xueqing et al. 2012. “Influenza Virus A/Beijing/501/2009(H1N1) NS1 Interacts with β-Tubulin and Induces Disruption of the Microtubule Network and Apoptosis on A549 Cells” ed. Michael C. W. Chan. PLoS ONE 7(11): e48340. https://dx.plos.org/10.1371/journal.pone.0048340 (November 2, 2020).
42.  ,
,  et al. 2000. “Eukaryotic Translation Initiation Factor 4GI Is a Cellular Target for NS1 Protein, a Translational Activator of Influenza Virus.” Molecular and Cellular Biology 20(17): 6259-68. http://mcb.asm.org/ (November 3, 2020).
et al. 2000. “Eukaryotic Translation Initiation Factor 4GI Is a Cellular Target for NS1 Protein, a Translational Activator of Influenza Virus.” Molecular and Cellular Biology 20(17): 6259-68. http://mcb.asm.org/ (November 3, 2020).
43. Burgui, Idoia, 
 , Juan
, Juan  , and Amelia Nieto. 2003. “PABP1 and EIF4GI Associate with Influenza Virus NS1 Protein in Viral MRNA Translation Initiation Complexes.” Journal of General Virology 84(12): 3263-74. https://www.microbiologyresearch.org/content/journal/jgv/10.1099/vir.0.19487-0 (November 3, 2020).
, and Amelia Nieto. 2003. “PABP1 and EIF4GI Associate with Influenza Virus NS1 Protein in Viral MRNA Translation Initiation Complexes.” Journal of General Virology 84(12): 3263-74. https://www.microbiologyresearch.org/content/journal/jgv/10.1099/vir.0.19487-0 (November 3, 2020).
44.  , Ana
, Ana  et al. 1999. “Interaction of Influenza Virus NS1 Protein and the Human Homologue of Staufen in Vivo and in Vitro.” Nucleic Acids Research 27(11): 2241-47. https://academic.oup.com/nar/article/27/11/2241/1252689 (November 3, 2020).
et al. 1999. “Interaction of Influenza Virus NS1 Protein and the Human Homologue of Staufen in Vivo and in Vitro.” Nucleic Acids Research 27(11): 2241-47. https://academic.oup.com/nar/article/27/11/2241/1252689 (November 3, 2020).
45. MDCK клетка-продуцент белков вируса гриппа (варианты): патент RU2681482, Российская Федерация, заявка RU2018105364, заявл. 13.02.2018, опубл. 06.03.2019.
46. Laemmli U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4 //nature. - 1970. - Т. 227. - №. 5259. - С. 680-685.
47. Shaldzhyan A. et al. Clean and folded: Production of active, high quality recombinant human interferon-λ1 //Process Biochemistry. - 2021. - Т. 111. - С. 32-39.
48. Кривицкая В.З. et al. Получение и характеристика панели моноклональных антител, специфичных к белку NS1 вируса гриппа А // Биотехнология. 2017. Vol. 33, № 5. P. 61-72.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> федеральное государственное автономное образовательное
учреждение высшего образования «Санкт-Петербургский
политехнический университет Петра Великого»
<120> Штамм бактерий Escherichia coli — продуцент рекомбинантного белка NS1
<160> 3
<170> PatentIn version 3.5
<210> 1
<211> 693
<212> DNA
<213> Escherichia coli
<400> 1
atggatccaaacactgtgtcaagctttcaggtagattgctttctttggcatgtccgcaaacgagttgcagaccaa
gaactaggtgatgccccattccttgatcggcttcgccgagatcagaaatccctaagaggaaggggcagcaccctc
ggtctggacatcgagacagccacacgtgctggaaagcagatagtggagcggattctgaaagaagaatccgatgag
gcacttaaaatgaccatggcctctgtacctgcgtcgcgttacctaactgacatgactcttgaggaaatgtcaagg
gagtggtccatgctcatacccaagcagaaagtggcaggccctctttgtatcagaatggaccaggcgatcatggat
aagaacatcatactgaaagcgaacttcagtgtgatttttgaccggctggagactctaatattgctaagggctttc
accgaagagggagcaattgttggcgaaatttcaccattgccttctcttccaggacatactgctgaggatgtcaaa
aatgcagttggagtcctcatcgggggacttgaatggaatgataacacagttcgagtctctgaaactctacagaga
ttcgcttggagaagcagtaatgagaatgggagacctccactcactccaaaacagaaacgagaaatggcgggaaca
attaggtcagaagtttga
<210> 2
<211> 182
<212> PRT
<213> Escherichia coli
<400> 2
MetAspProAsnThrValSerSerPheGlnValAspCysPheLeuTrpHisValArgLysArgValAlaAspGln
GluLeuGlyAspAlaProPheLeuAspArgLeuArgArgAspGlnLysSerLeuArgGlyArgGlySerThrLeu
GlyLeuAspIleGluThrAlaThrArgAlaGlyLysGlnIleValGluArgIleLeuLysGluGluSerAspGlu
AlaLeuLysMetThrMetAlaSerValProAlaSerArgTyrLeuThrAspMetThrLeuGluGluMetSerArg
GluTrpSerMetLeuIleProLysGlnLysValAlaGlyProLeuCysIleArgMetAspGlnAlaIleMetAsp
LysAsnIleIleLeuLysAlaAsnPheSerValIlePheAspArgLeuGluThrLeuIleLeuLeuArgAlaPhe
ThrGluGluGlyAlaIleValGlyGluIleSerProLeuProSerLeuProGlyHisThrAlaGluAspValLys
AsnAlaValGlyValLeuIleGlyGlyLeuGluTrpAsnAspAsnThrValArgValSerGluThrLeuGlnArg
PheAlaTrpArgSerSerAsnGluAsnGlyArgProProLeuThrProLysGlnLysArgGluMetAlaGlyThr
IleArgSerGluValHisHisHisHisHisHis
<210> 3
<211> 28
<212> PRT
<213> artificial sequence
AACCTTCATATggATCCAAACACTgTg
<210> 4
<211> 30
<212> PRT
<213> artificial sequence
TTAACTCgAgAACTTCTgACCTAATTgTTC
<---
Изобретение относится к биотехнологии. Предложена рекомбинантная плазмидная ДНК pET22b(+)-NS1-6×His для экспрессии в клетках Escherichia coli рекомбинантного неструктурного белка NS1 вируса гриппа А. Предложен также штамм Escherichia coli BL-21(DE3)-pET22b(+)-NS1, являющийся продуцентом рекомбинантного неструктурного белка NS1 вируса гриппа А и полученный путем трансформации клеток Escherichia coli BL-21(DE3) рекомбинантной плазмидной ДНК pET22b(+)-NS1-6×His. Указанный штамм депонирован в Биоресурсном Центре Всероссийской Коллекции Промышленных Микроорганизмов (БРЦ ВКПМ) НИЦ «Курчатовский институт» ГосНИИгенетика под № В-14117. Изобретение позволяет получить стабильный при хранении рекомбинантный неструктурный белок NS1 гриппа А, который может быть использован в качестве мишени при разработке подходов направленной противовирусной терапии. 2 н.п. ф-лы, 6 ил.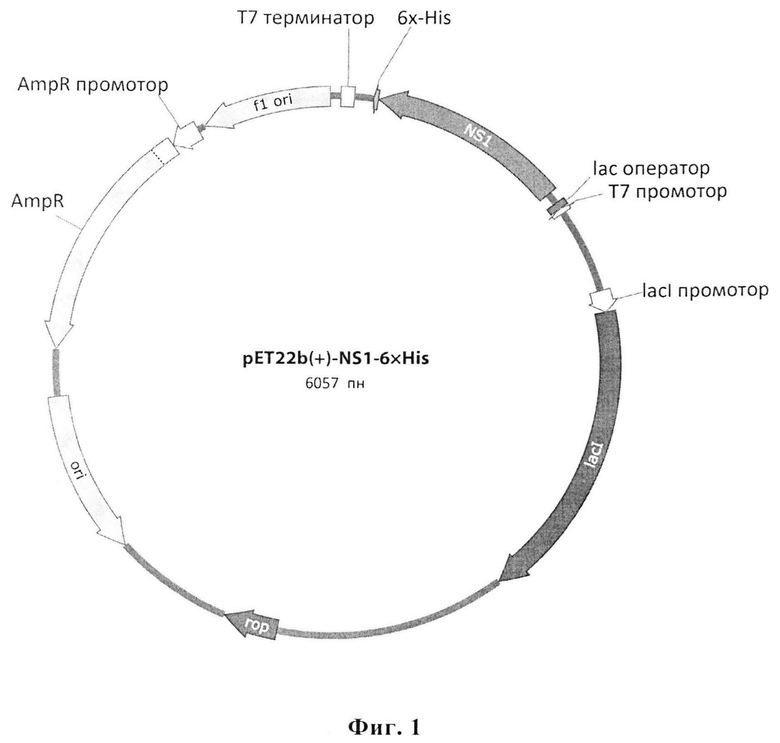
1. Рекомбинантная плазмидная ДНК pET22b(+)-NS1-6×His для экспрессии в клетках Е. coli рекомбинантного неструктурного белка NS1 вируса гриппа А, имеющего последовательность SEQ ID NO: 2, имеющая карту плазмиды, как показано на Фиг. 1, включающая следующие участки:
- ген β-лактамазы (AmpR), определяющий устойчивость бактериальных клеток к ампициллину;
- участок инициации репликации (ori);
- ROP ген, регулирующий копийность плазмиды;
- фрагмент, содержащий Т7 терминатор транскрипции;
- транскрипционный репрессор LacI, регулирующий транскрипцию T7lac-промотора;
- NdeI/XhoI фрагмент, содержащий искусственный ген NS1, в котором нуклеотидная последовательность соответствует SEQ ID NO: l и который кодирует рекомбинантный белок NS1, имеющий аминокислотную последовательность SEQ ID NO: 2;
- AmpR промотор;
- LacI промотор;
- Т7 промотор;
- lac оператор;
- 6×His.
2. Штамм Escherichia coli BL-21(DE3)-pET22b(+)-NS1 - продуцент рекомбинантного неструктурного белка NS1 вируса гриппа А, имеющего последовательность SEQ ID NO: 2, и депонированный в Биоресурсном Центре Всероссийской Коллекции Промышленных Микроорганизмов (БРЦ ВКПМ) НИЦ «Курчатовский институт» - ГосНИИгенетика под № В-14117, при этом штамм получен путем трансформации клеток штамма Е. coli BL-21(DE3) рекомбинантной плазмидной ДНК pET22b(+)-NS1-6×His по п. 1.
| MARC D | |||
| ET AL | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Nucleic Acids Res | |||
| Многоступенчатая активно-реактивная турбина | 1924 |
|
SU2013A1 |
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| Найдено онлайн: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3592425/ Дата обращения 15.08.2022 | |||
| SADEGHI M | |||
Авторы
Даты
2023-02-07—Публикация
2021-12-24—Подача