Изобретение относится к области генной инженерии и биотехнологии, а именно к направляющим РНК, которые могут быть использованы в системах CRISPR-Cas12 в составе рибонуклеопротеиновых комплексов для выявления (обнаружения, детекции) гена антибиотикоустойчивости blа-NDM-1, а также к способам получения препаратов рибонуклеопротеинового комплекса CRISPR-Cas и к самим препаратам.
Изобретение позволяет in vitro выявлять единичные копии гена антибиотикоустойчивости bla-NDM-1.
Направляющие РНК, описанные в настоящей заявке, могут быть использованы для детекции гена антибиотикоустойчивости bla-NDM-1 после проведения специфической амплификации фрагмента гена антибиотикоустойчивости bla-NDM-1. Амплификация при этом может быть проведена различными способами, среди которых полимеразная цепная реакция (PCR); петлевая изотермическая амплификация (LAMP); геликаза-зависимая амплификация (HDA); рекомбиназа-опосредованная амплификация (RPA); амплификация со смещением цепи (SDA); амплификация, основанная на последовательности нуклеиновых кислот (NASBA); опосредованная транскрипцией амплификация (ТМА); амплификация, опосредованная никирующим ферментом (NEAR); круговая амплификация (RCA) и многие другие виды амплификации.
Направляющие РНК, описанные в настоящей заявке, могут быть использованы для разработки высокочувствительных и высокотехнологичных диагностических систем нового поколения на основе CRISPR технологий для совершенствования методов диагностики инфекционных заболеваний.
Для решения эпидемиологических задач по расшифровке вспышек инфекционных болезней, выявления и идентификации возбудителя, а также детекции специфических бактериальных генов необходимы разработка и внедрение в практику работы надзорных и мониторинговых служб современных технологий молекулярной эпидемиологии. Одной из таких технологий является использование элементов генетического редактирования системы CRISPR-Cas. Данная технология развивается достаточно эффективно в отношении создания средств лечения некоторых болезней, несмотря на ряд трудностей, связанных с возникновением непредвиденных мутаций. При углубленных исследованиях в области применения CRISPR-Cas системы, было выяснено, что она может быть использована для тонких диагностических процедур при выявлении возбудителя/ей инфекции у человека, а также их генотипирования.
В 2018 году было показано, что один из ферментов CRISPR системы - Cas12 после распознавания своей целевой ДНК-мишени начинает неспецифически гидролизовать одноцепочечную ДНК. Такое свойство Cas12 можно использовать в качестве индикатора присутствия определенной мишени, например, генома вируса или бактерии. Исследователи использовали это открытие для создания технологической платформы обнаружения нуклеиновых кислот, известной как DETECTR (DNA Endonuclease Targeted CRISPR Trans Reporter - ДНК-нацеленная эндонуклеаза CRISPR транс репортер). Впервые DETECTR была использована для выявления и генотипирования вируса папилломы человека (HPV). Предложенная платформа объединяет нуклеазу Cas12a, ее направляющую РНК, специфичную к нуклеиновой кислоте HPV, флуоресцентную репортерную молекулу. Технология DETECTR используется для обнаружения целевой ДНК-мишени после предварительной амплификации [J.S. Chen, Е. Ma, L.B. Harrington, М. Da Costa, X. Tian, J.M. Palefsky, J. A. Doudna, CRISPR-Cas 12a target binding unleashes indiscriminate single-stranded DNase activity, Science 360(6387) (2018) 436-439].
He менее важным приложением системы CRISPR-Cas является идентификация бактериальных патогенов и детекция специфических бактериальных генов. Так, например, с помощью платформы SHERLOCK удалось корректно генотипировать ряд штаммов Escherichia coli и Pseudomonas aeruginosa при низкой перекрестной реактивности. Кроме того, платформа SHERLOCK использована для дифференциации клинических изолятов Klebsiella pneumoniae с двумя различными генами антибиотикоустойчивости, что открывает значительные перспективы к созданию мультиплексных систем для одновременной идентификации бактеральных патогенов и выявления у них генов антибиотикоустойчивости.
В связи с этим крайне актуальной является задача разработки новых эффективных методик выявления генов антибиотикоустойчивости у бактериальных патогенов, основанных на генетических технологиях, таких как CRISPR-Cas.
Из уровня техники известны научные статьи, описывающие обнаружение гена антибиотикоустойчивости bla-NDM-1 в биологическом материале [Shin J, Kim SR, Xie Z, Jin YS, Wang YC. A CRISPR/Cas12a-Based System for Sensitive Detection of Antimicrobial-Resistant Genes in Carbapensm-ResistantEnterobacterales. Biosensors (Basel). 2024; 14(4): 194. Published 2024 Apr 16. doi:10.3390/bios 14040194; Xu H, Tang H, Li R, et al. A New Method Based on LAMP-CRISPR-Cas 12a-Lateral Flow Immunochromatographic Strip for Detection. Infect Drug Resist. 2022; 15:685-696. Published 2022 Feb 27. doi:10.2147/IDR.S348456]. Однако, предел обнаружения гена антибиотикоустойчивости bla-NDM-1 в приведенных публикациях в пересчете на количество копий ДНК в реакции достаточно низкий и составляет 2,7-2,9×109 копий/реакция и 6×103 копий/реакция, соответственно [Shin J, Kim SR, Xie Z, Jin YS, Wang YC. A CRISPR/Cas12a-Based System for Sensitive Detection of Antimicrobial-Resistant Genes in Carbapenem-Resistant Enterobacterales. Biosensors (Basel). 2024;14(4):194. Published 2024 Apr 16. doi:10.3390/bios 14040194; Xu H, Tang H, Li R, et al. A New Method Based on LAMP-CRISPR-Cas 12a-Lateral Flow Immunochromatographic Strip for Detection. Infect Drug Resist. 2022;15:685-696. Published 2022 Feb 27. doi:10.2147/IDR.S348456].
До сих пор большинство наборов для обнаружения бета-лактамаз расширенного спектра, к которым в том числе относится бета-лактамаза, кодируемая геном bla-NDM-1, основаны на методологии диффузионного теста в агаре и определении изоэлектрической точки, которая обычно считается достаточной для идентификации штаммов, продуцирующих бета-лактамазы расширенного спектра. Однако из-за появления большого количества различных бета-лактамаз bla-SHV, bla-TEM и bla-CTX-M изоэлектрофокусирование, по-видимому, не является подходящим методом для установления фенотипа бета-лактамаз расширенного спектра. Кроме того, описанные методы требуют продолжительного времени, специализированного оборудования, а также квалифицированного персонала (микробиология).
Ближайшими аналогами заявляемого решения являются изобретения по патентам RU 2745637 (дата приоритета 15.04.2020) направленное на получение направляющих РНК, предназначенных для разработки высокочувствительных и высокотехнологичных диагностических систем нового поколения для выявления гена антибиотикоустойчивости blaVIM-2 (металло-бета-лактамаза класс В VIM-2) Pseudomonas aeruginosa; RU 2782315 (дата приоритета 27.12.2021), раскрывающее техническую возможность получения направляющих РНК, предназначенных для разработки высокочувствительных и высокотехнологичных диагностических систем нового поколения для выявления гена антибиотикоустойчивости mec A Staphylococcus aureus на основе CRISPR технологий; RU 2782739 (дата приоритета 27.12.2021), раскрывающее возможность получения направляющих РНК, предназначенных для разработки высокочувствительных и высокотехнологичных диагностических систем нового поколения для выявления exoU, кодирующего экзотоксин системы секреции третьего типа, Pseudomonas aeruginosa на основе CRISPR технологий. Однако, для расширения спектра подобных диагностических систем и совершенствования методов диагностики инфекционных заболеваний, эпидемиологического надзора и контроля за распространением антибиотикоустойчивых микроорганизмов необходимо разрабатывать новые методы для выявления генов антибиотикоустойчивости патогенных микроорганизмов.
Исходя из вышеизложенного, возникает техническая проблема, заключающаяся в необходимости разработки и получения направляющих РНК для выявления единичных копий гена антибиотикоустойчивости bla-NDM-1, in vitro.
Предложенная технология перспективна для разнообразных применений, включая количественное определение ДНК/РНК, быструю мультиплексную детекцию экспрессии, другие виды чувствительной детекции, например, выявление загрязнения образцов нуклеиновыми кислотами. Технология, основанная на CRISPR-Cas, является многофункциональной, устойчивой к ошибкам технологией детекции ДНК, пригодной для быстрой постановки диагнозов, включая инфекционные заболевания, и генотипирования инфекционных агентов и выявление генов антибиотикоустойчивости и генов, кодирующих экзотоксины, бактериальных патогенов.
Применение предложенной технологии делает возможным создание диагностических систем нового поколения, которые будут обладать следующими свойствами:
высокая чувствительность;
возможность проведения диагностики у постели больного;
возможность проведения диагностики в полевых условиях без применения специализированного высокотехнологичного оборудования;
скорость и простота анализа;
сниженная стоимость анализа;
отсутствие необходимости оснащения диагностической лаборатории дорогостоящим оборудованием;
отсутствие необходимости проведения выделения нуклеиновых кислот возбудителя.
Изобретение относится к новым средствам - направляющим РНК, которые могут быть использованы в системах CRISPR-Cas12 для ультрачувствительного выявления, идентификации, обнаружения или детекции гена антибиотикоустойчивости bla-NDM-1 в биологических образцах.
Технической задачей предложенного изобретения является разработка новых средств - направляющих РНК, которые могут быть использованы в системах CRISPR-Cas12 с белками Cas12, например, LbCpf1 из Lachnospiraceae, для ультрачувствительного выявления гена антибиотикоустойчивости bla-NDM-1.
При осуществлении настоящего изобретения, согласно приведенной в формуле изобретения совокупности существенных признаков, достигается неожиданный технический результат возможность ультрачувствительного выявления гена антибиотикоустойчивости bla-NDM-1 до единичных копий в одной реакции. Изобретение обеспечивает повышение эффективности выявления гена антибиотикоустойчивости bla-NDM-1 минимум в 5 раз. Также предложенное изобретение позволяет ускорить и упростить получение финального препарата рибонуклеопротеинового комплекса CRISPR-Cas.
Технический результат достигается за счет:
разработки молекул направляющих РНК, которые могут быть использованы в системах CRISPR-Cas 12 для ультра чувствительного выявления гена антибиотикоустойчивости bla-NDM-1, где указанные направляющие РНК выбраны из последовательностей SEQ ID NO: 1-8, способны связываться с целевыми высоко консервативными участками гена антибиотикоустойчивости bla-NDM-1, содержат РНК-шпильку, которая распознается РНК-направляемой ДНК-эндонуклеазой LbCpf1 из Lachnospiraceae, с обеспечением выявления единичных копий гена антибиотикоустойчивости bla-NDM-1.
применения РНК-направляемой ДНК-эндонуклеазы LbCpf1 из Lachnospiraceae, полученной согласно способу, разработанному авторами ранее (Патент RU №2707542, дата приоритетат 28.03.2019), для создания рибонуклеопротеиновых комплексов (РПК) системы CRISPR-Cas, пригодных для детекции гена антибиотикоустойчивости bla-NDM-1 в ультра низких концентрациях (единичные копии).
разработки набора специфических олигонуклеотидов, выбранных из SEQ ID NO: 9-14, для предварительной амплификации фрагмента гена антибиотикоустойчивости bla-NDM-1.
оптимизации условий проведения предварительной амплификации фрагмента гена антибиотикоустойчивости bla-NDM-1.
определения условий проведения ультрачувствительной детекции гена антибиотикоустойчивости bla-NDM-1 и установления последовательности стадий метода.
Направляющие РНК, согласно настоящему изобретению, соответствуют высоко консервативным фрагментам гена антибиотикоустойчивости bla-NDM-1. Наиболее предпочтительны направляющие РНК, распознающиеся РНК-направляемой ДНК-эндонуклеазой LbCpf1 из Lachnospiraceae, характеризующиеся, имеющие или содержащие нуклеотидную последовательность, выбранную из:
SEQ ID NO: l;
SEQ ID NO: 2;
SEQ ID NO: 3;
SEQ ID NO: 4;
SEQ ID NO: 5;
SEQ ID NO: 6;
SEQ ID NO: 7;
SEQ ID NO: 8;
или идентичной любой из них по меньшей мере на 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99%,
или комплементарной любой из них,
или гибридизующейся с любой из них в строгих условиях. Специфические олигонуклеотиды для проведения предварительной амплификации
фрагмента гена антибиотикоустойчивости bla-NDM-1, согласно настоящему изобретению, соответствуют высоко консервативному участку гена антибиотикоустойчивости bla-NDM-1. Наиболее предпочтительны олигонуклеотиды, характеризующиеся, имеющие или содержащие нуклеотидную последовательность, выбранную из:
SEQ ID NO: 9;
SEQ ID NO: 10;
SEQ ID NO: 11;
SEQ ID NO: 12;
SEQ ID NO: 13;
SEQ ID NO: 14;
или идентичной любой из них по меньшей мере на 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99%,
или комплементарной любой из них,
или гибридизующейся с любой из них в строгих условиях.
Согласно предложенному изобретению, получают рибонуклеопротеиновые комплексы (РПК), состоящие из по меньшей мере одной направляющей РНК и РНК-направляемой ДНК-нуклеазы системы CRISPR-Cas LbCpf1 из Lachnospiraceae, пригодные для использования для выявления гена антибиотикоустойчивости bla-NDM-1 в ультранизких концентрациях (единичные копии).
Препараты РПК представляют собой растворы, содержащие направляющую РНК, выбранную из SEQ ID NO: 1-8, объединенную с белком системы CRISPR-Cas (LbCpf1 из Lachnospiraceae) или лиофильно высушенные РПК.
Полученные направляющие РНК могут быть использованы в составе набора для обнаружения гена антибиотикоустойчивости bla-NDM-1 с инструкцией по применению.
Набор может дополнительно включать компоненты для проведения предварительной амплификации высококонсервативного фрагмента гена антибиотикоустойчивости bla-NDM-1, в том числе один или несколько специфических олигонуклеотидов, выбранных из SEQ ID NO: 9-14. При этом по меньшей мере одна направляющая РНК в составе набора может находиться в комплексе с белком системы CRISPR-Cas (LbCpf1 из Lachnospiraceae) в одном контейнере или отдельно в разных контейнерах.
Способ получения препарата рибонуклеопротеинового комплекса CRISPR-Cas предусматривает:
(i) объединение Cas-белка семейства CRISPR-Cas LbCpf1 из Lachnospiraceae, в комплекс с по меньшей мере одной направляющей РНК с SEQ ID NO: 1-8 и при необходимости
(ii) лиофильную сушку рибонуклеопротеинового комплекса CRISPR-Cas, полученного на стадии (i),
с получением, таким образом, препарата рибонуклеопротеинового комплекса CRISPR-Cas.
Предложен препарат рибонуклеопротеинового комплекса CRISPR-Cas для выявления гена антибиотикоустойчивости bla-NDM-1, который может быть получен раскрытым в настоящей заявке способом. Препарат содержит Cas-белок семейства CRISPR-Cas LbCpf1 из Lachnospiraceae в комплексе с по меньшей мере одной направляющей РНК с SEQ ID NO: 1-8.
Препарат может быть представлен как в жидкой форме - раствора указанного рибонуклеопротеинового комплекса CRISPR-Cas, так и в виде лиофилизата - лиофильно высушенного порошка указанного рибонуклеопротеинового комплекса CRISPR-Cas.
Предложенная технология позволяет определить единичные копии гена антибиотикоустойчивости bla-NDM-1 в биологических образцах пациента, выбранных из жидкости и/или ткани, предположительно содержащих ген антибиотикоустойчивости bla-NDM-1. Биологическим образцом может быть образец крови, сыворотки или плазмы крови, клеток крови, слюны, мокроты, лимфоидных тканей, тканей кроветворных органов и других биологических материалов от пациента, которые могут быть использованы для анализа на наличие гена антибиотикоустойчивости bla-NDM-1.
Краткое описание чертежей
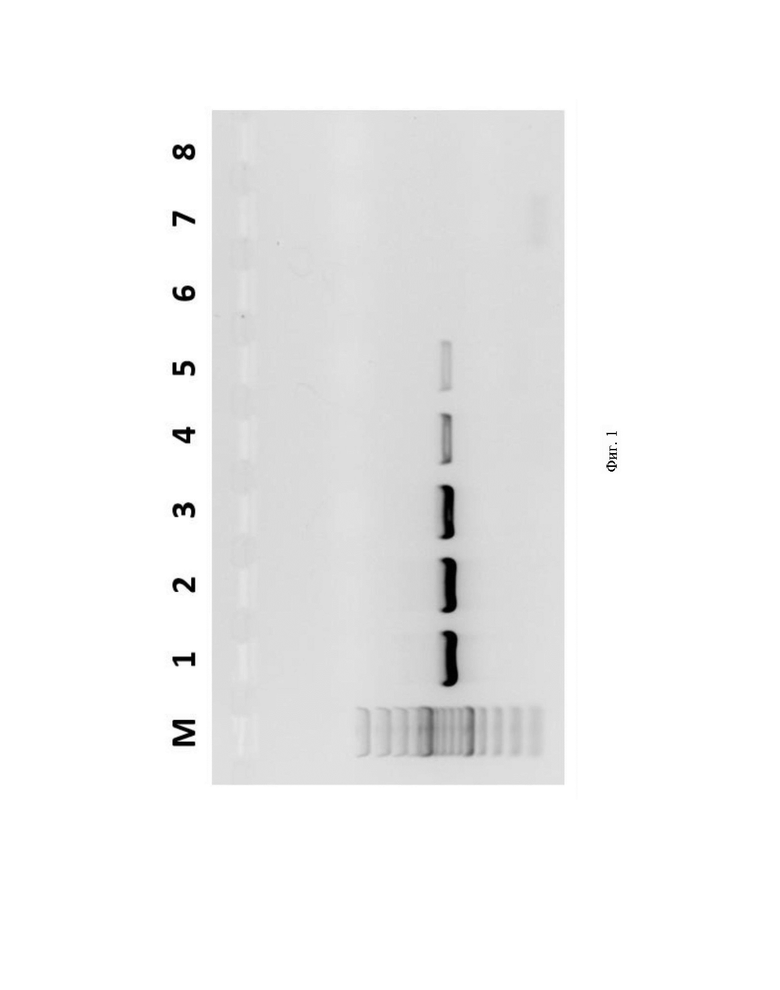
Фиг.1. Визуализация амплифицированного фрагмента гена антибиотикоустойчивости bla-NDM-1 (размером 680 п. о.) после предварительной амплификации с использованием олигонуклеотидов NDM-1 for 1 и NDM-1 rev 1 при помощи электрофореза в агарозном геле, где цифрами 1-8 обозначены:
1 - Продукт, полученный в ходе амплификации 3,48×106 копий модельной матрицы pGEM-T-bla-NDM-1;
2 Продукт, полученный в ходе амплификации 3,48×105 копий модельной матрицы pGEM-T-bla-NDM-1;
3 - Продукт, полученный в ходе амплификации 3,48×104 копий модельной матрицы pGEM-T-bla-NDM-1;
4 Продукт, полученный в ходе амплификации 3480 копий модельной матрицы pGEM-T-bla-NDM-1;
5 Продукт, полученный в ходе амплификации 348 копий модельной матрицы pGEM-T-bla-NDM-1;
6 - Продукт, полученный в ходе амплификации 34,8 копии модельной матрицы pGEM-T-bla-NDM-1;
7 Продукт, полученный в ходе амплификации 3,48 копии модельной матрицы pGEM-T-bla-NDM-1;
8 - отрицательный контроль, не содержащий модельной матрицы pGEM-T-bla-NDM-1;
М - стандарты молекулярных масс: снизу-вверх 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000, 1200, 1500, 2000, 3000 пар нуклеотидов (GeneRuler 100 bp Plus, Thermo Fisher Scientific, США).
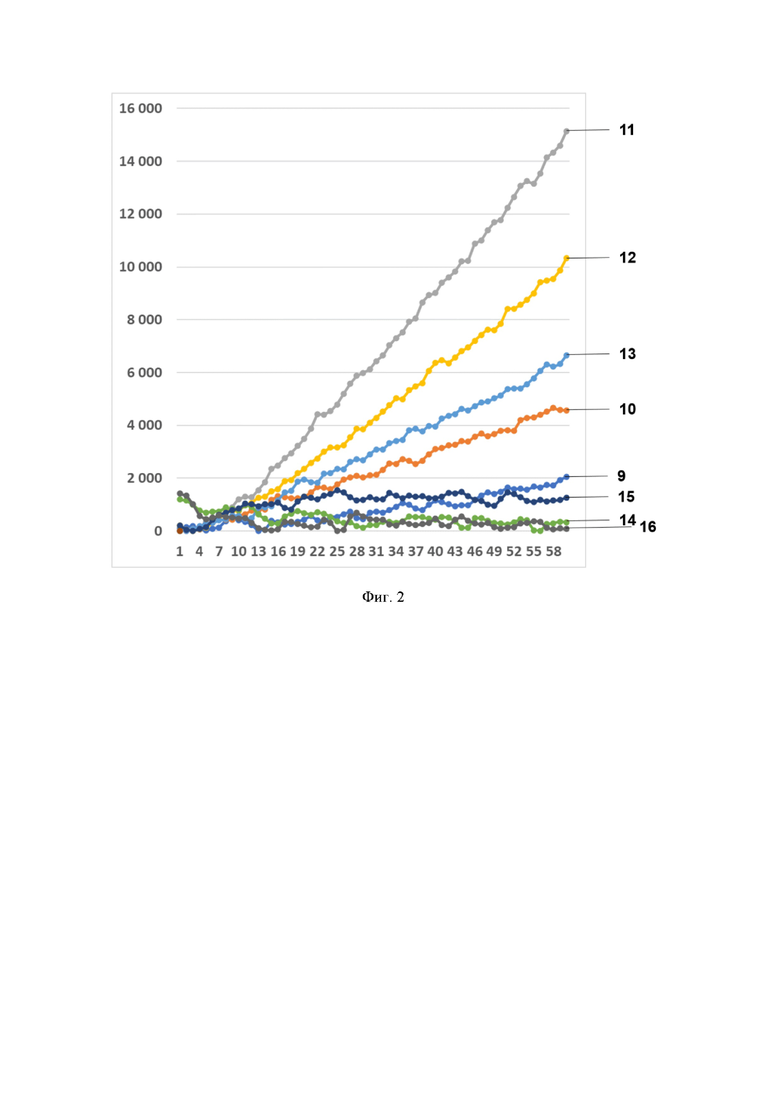
Фиг. 2. Профиль флуоресценции в реальном времени для предварительно амплифицированного фрагмента гена антибиотикоустойчивости bla-NDM-1, обработанного рибонуклеопортеиновым комплексом, содержащим направляющую РНК crRNA blaNDM №100 и белок LbCpf1, на котором посредством графика визуализировано выявление фрагмента модельной матрицы pGEM-T-bla-NDM-1 с помощью рибонуклеопротеиновых комплексов LbCpf1 из Lachnospiraceae, содержащих направляющую РНК crRNA blaNDM №100, где:
- по вертикали указаны значения нормализированного флуоресцентного сигнала, создаваемого красителем;
- по горизонтали указано время флуоресценции, в минутах;
- на кривых 9-16 показано количество копий нуклеиновой кислоты на реакцию, а именно:
9 - 3480000 копий/реакция;
10 - 348000 копий/реакция;
11 - 34800 копий/реакция;
12 - 3480 копий/реакция;
13 - 348 копий/реакция;
14 34,8 копий/реакция;
15 - 3,48 копий/реакция;
16 - К- (отрицательный контроль).
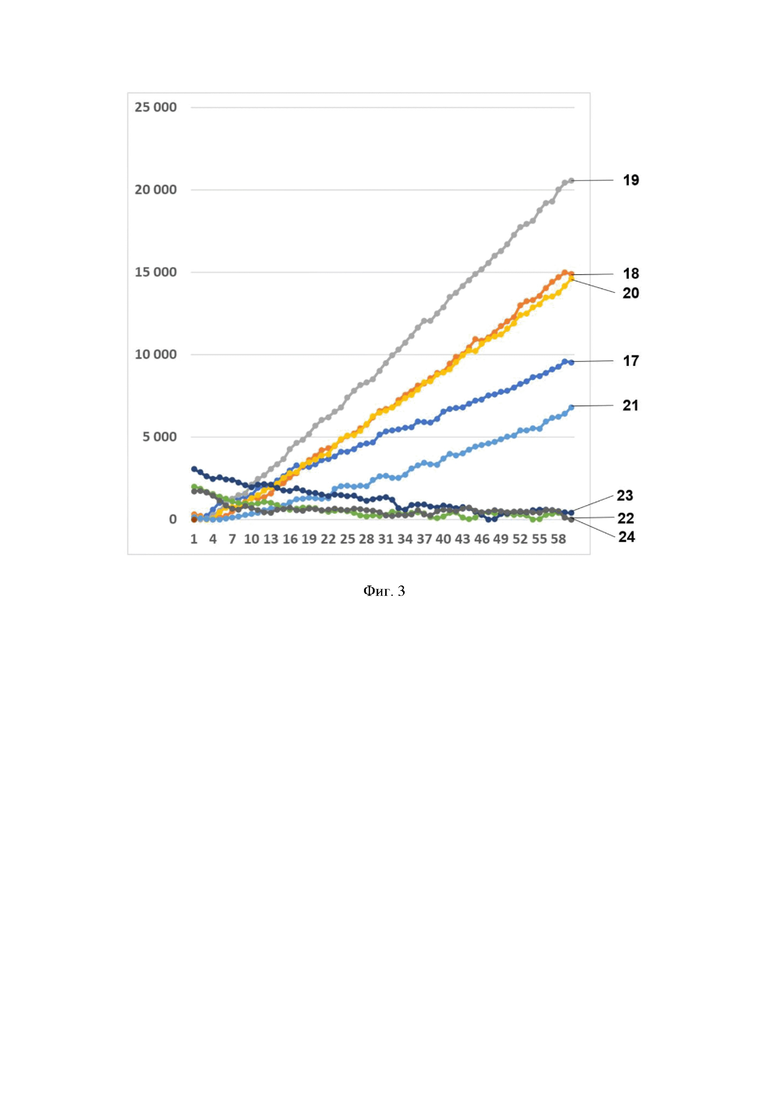
Фиг. 3. Профиль флуоресценции в реальном времени для предварительно амплифицированного фрагмента гена антибиотикоустойчивости bla-NDM-1, обработанного рибонуклеопортеиновым комплексом, содержащим направляющую РНК crRNA blaNDM №124 и белок LbCpf1, на котором посредством графика визуализировано выявление фрагмента модельной матрицы pGEM-T-bla-NDM-1 с помощью рибонуклеопротеиновых комплексов LbCpf1 из Lachnospiraceae, содержащих направляющую РНК crRNA blaNDM №124, где:
- по вертикали указаны значения нормализированного флуоресцентного сигнала, создаваемого красителем;
- по горизонтали указано время флуоресценции, в минутах;
- на кривых 17-24 показано количество копий нуклеиновой кислоты на реакцию, а именно:
17 - 3480000 копий/реакция;
18 - 348000 копий/реакция;
19 - 34800 копий/реакция;
20 - 3480 копий/реакция;
21 - 348 копий/реакция; 22-34,8 копий/реакция;
23 - 3,48 копий/реакция;
24 - К- (отрицательный контроль).
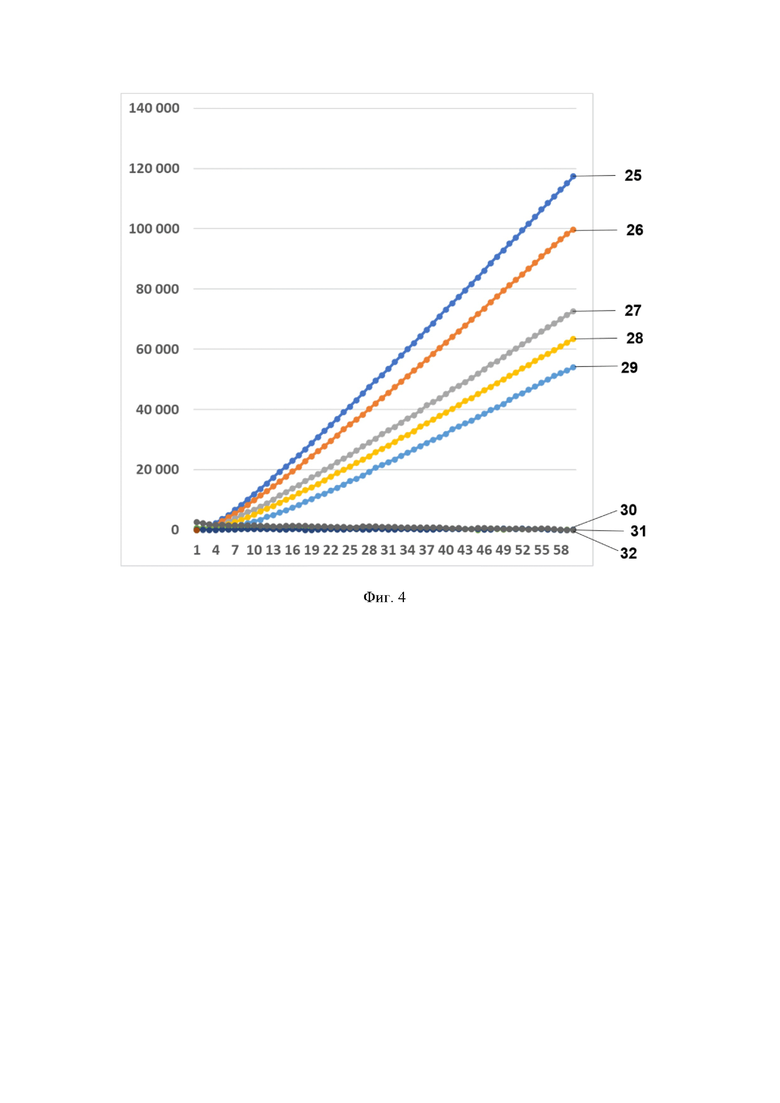
Фиг. 4. Профиль флуоресценции в реальном времени для предварительно амплифицированного фрагмента гена антибиотикоустойчивости bla-NDM-1, обработанного рибонуклеопортеиновым комплексом, содержащим направляющую РНК crRNA blaNDM №204 и белок LbCpf1, на котором посредством графика визуализировано выявление фрагмента модельной матрицы pGEM-T-bla-NDM-1 с помощью рибонуклеопротеиновых комплексов LbCpf1 из Lachnospiraceae, содержащих направляющую РНК crRNA blaNDM №204, где:
- по вертикали указаны значения нормализированного флуоресцентного сигнала, создаваемого красителем;
- по горизонтали указано время флуоресценции, в минутах;
- на кривых 25 - 32 показано количество копий нуклеиновой кислоты на реакцию, а именно:
25 - 3480000 копий/реакция;
26 - 348000 копий/реакция;
27 - 34800 копий/реакция;
28 - 3480 копий/реакция;
29 - 348 копий/реакция;
30 - 34,8 копий/реакция;
31 - 3,48 копий/реакция;
32- К- (отрицательный контроль).
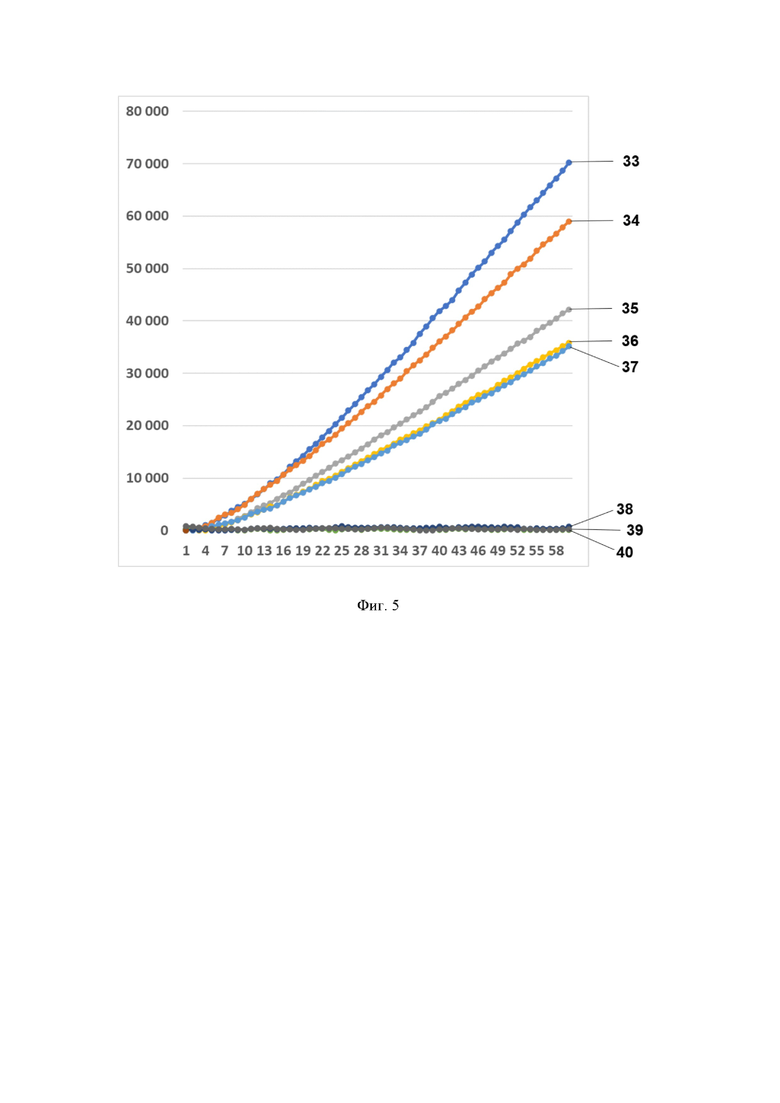
Фиг. 5. Профиль флуоресценции в реальном времени для предварительно амплифицированного фрагмента гена антибиотикоустойчивости bla-NDM-1, обработанного рибонуклеопортеиновым комплексом, содержащим направляющую РНК crRNA blaNDM №285 и белок LbCpf1, на котором посредством графика визуализировано выявление фрагмента модельной матрицы pGEM-T-bla-NDM-1 с помощью рибонуклеопротеиновых комплексов LbCpf1 из Lachnospiraceae, содержащих направляющую РНК crRNA blaNDM №285, где:
- по вертикали указаны значения нормализированного флуоресцентного сигнала, создаваемого красителем;
- по горизонтали указано время флуоресценции, в минутах;
- на кривых
33 - 40 показано количество копий нуклеиновой кислоты на реакцию, а именно:
33 - 3480000 копий/реакция;
34 - 348 000 копий/реакция;
35 - 34800 копий/реакция;
36 - 3480 копий/реакция;
37 - 348 копий/реакция;
38 - 34,8 копий/реакция;
39 - 3,48 копий/реакция;
40 - К- (отрицательный контроль).
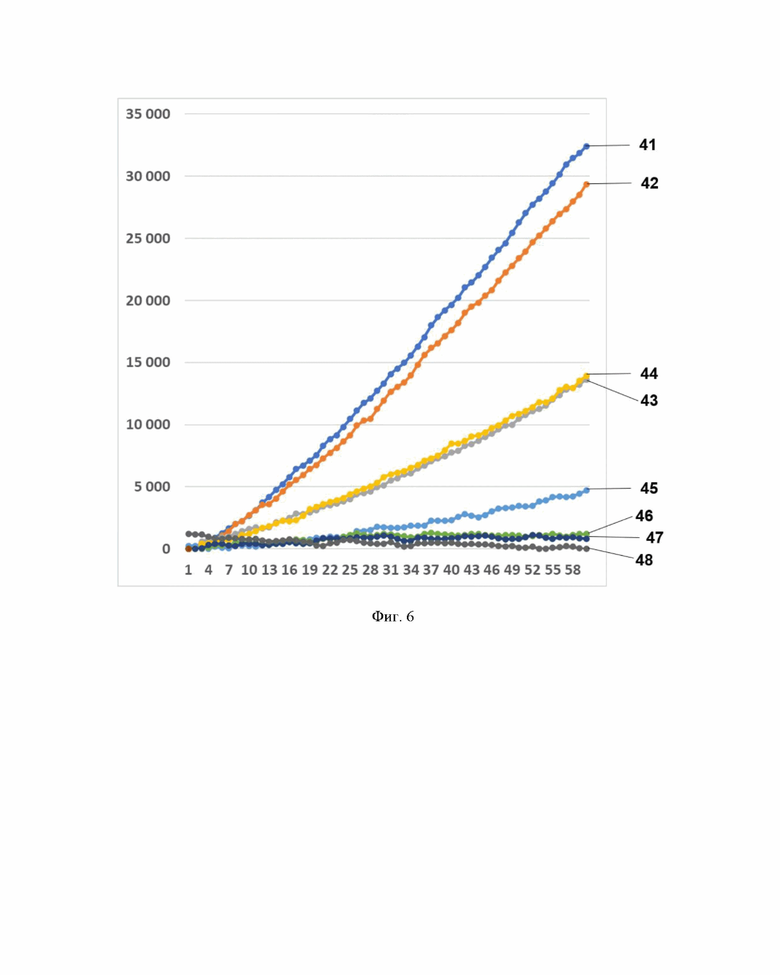
Фиг. 6. Профиль флуоресценции в реальном времени для предварительно амплифицированного фрагмента гена антибиотикоустойчивости bla-NDM-1, обработанного рибонуклеопортеиновым комплексом, содержащим направляющую РНК crRNA blaNDM №540 и белок LbCpf1, на котором посредством графика визуализировано выявление фрагмента модельной матрицы pGEM-T-bla-NDM-1 с помощью рибонуклеопротеиновых комплексов LbCpf1 из Lachnospiraceae, содержащих направляющую РНК crRNA blaNDM №540, где:
- по вертикали указаны значения нормализированного флуоресцентного сигнала, создаваемого красителем;
- по горизонтали указано время флуоресценции, в минутах;
- на кривых 41 - 48 показано количество копий нуклеиновой кислоты на реакцию, а именно:
41 - 3480000 копий/реакция;
42 - 348000 копий/реакция;
43 - 34800 копий/реакция;
44 - 3480 копий/реакция;
45 - 348 копий/реакция;
46 - 34,8 копий/реакция;
47 - 3,48 копий/реакция;
48 - К- (отрицательный контроль).
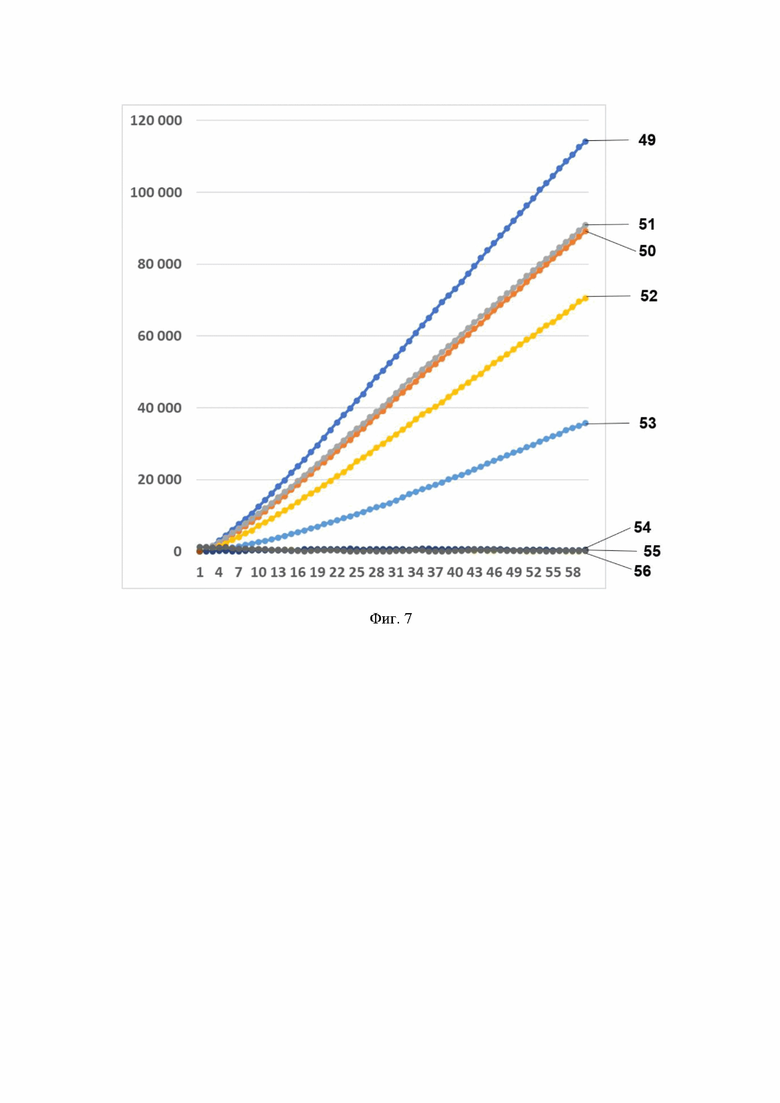
Фиг. 7. Профиль флуоресценции в реальном времени для предварительно амплифицированного фрагмента гена антибиотикоустойчивости bla-NDM-1, обработанного рибонуклеопортеиновым комплексом, содержащим направляющую РНК crRNA blaNDM №583 и белок LbCpf1, на котором посредством графика визуализировано выявление фрагмента модельной матрицы pGEM-T-bla-NDM-1 с помощью рибонуклеопротеиновых комплексов LbCpf1 из Lachnospiraceae, содержащих направляющую РНК crRNA blaNDM №583, где:
- по вертикали указаны значения нормализированного флуоресцентного сигнала, создаваемого красителем;
- по горизонтали указано время флуоресценции, в минутах;
- на кривых 49-56 показано количество копий нуклеиновой кислоты на реакцию, а именно:
49 - 3480000 копий/реакция;
50 - 348000 копий/реакция;
51 - 34800 копий/реакция;
52 - 3480 копий/реакция;
53 - 348 копий/реакция;
54 - 34,8 копий/реакция;
55 - 3,48 копий/реакция;
56 - К- (отрицательный контроль).
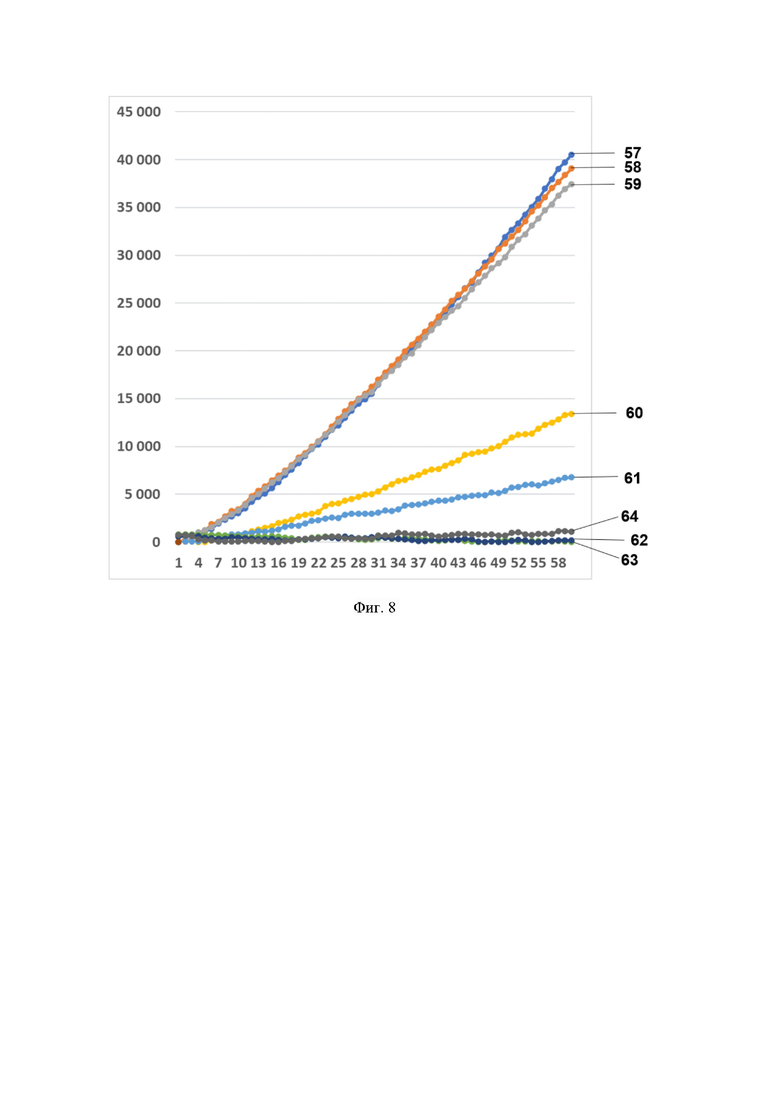
Фиг. 8. Профиль флуоресценции в реальном времени для предварительно амплифицированного фрагмента гена антибиотикоустойчивости bla-NDM-1, обработанного рибонуклеопортеиновым комплексом, содержащим направляющую РНК crRNA blaNDM №666 и белок LbCpf1, на котором посредством графика визуализировано выявление фрагмента модельной матрицы pGEM-T-bla-NDM-1 с помощью рибонуклеопротеиновых комплексов LbCpf1 из Lachnospiraceae, содержащих направляющую РНК crRNA blaNDM №666, где:
- по вертикали указаны значения нормализированного флуоресцентного сигнала, создаваемого красителем;
- по горизонтали указано время флуоресценции, в минутах;
- на кривых 57-64 показано количество копий нуклеиновой кислоты на реакцию, а именно:
57 - 3480000 копий/реакция;
58 - 348000 копий/реакция;
59 - 34800 копий/реакция;
60 - 3480 копий/реакция;
61 - 348 копий/реакция;
62 - 34,8 копий/реакция;
63 - 3,48 копий/реакция;
64 - К- (отрицательный контроль).
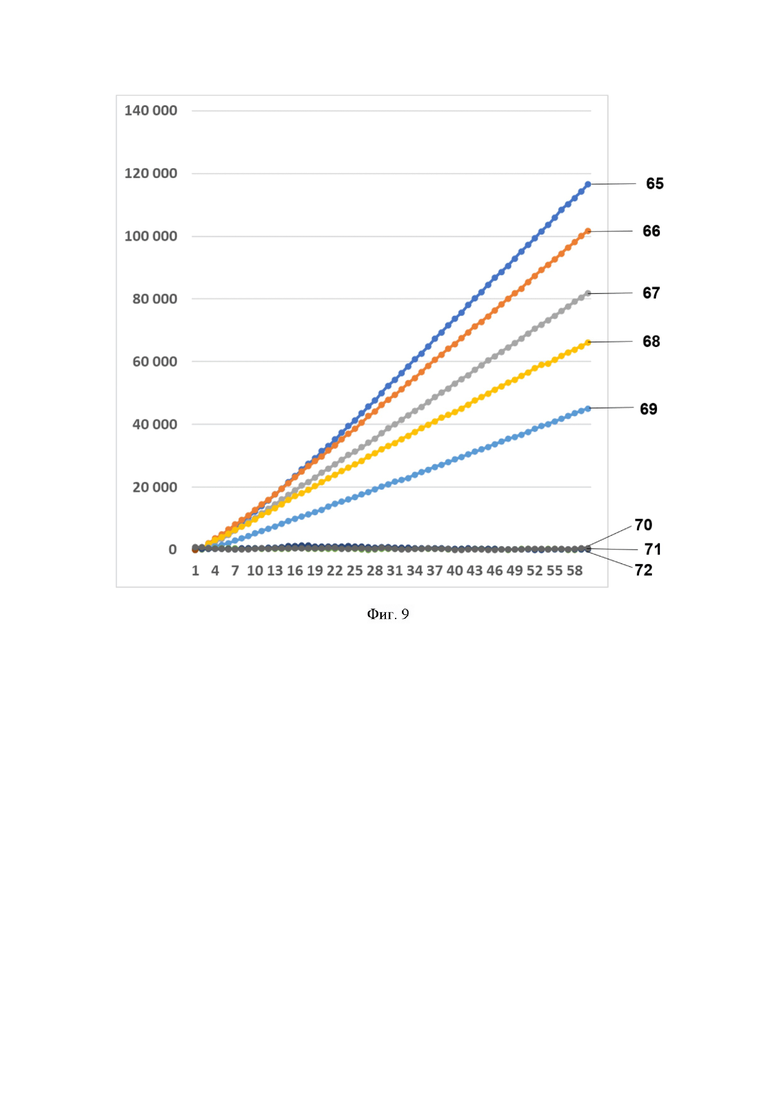
Фиг. 9. Профиль флуоресценции в реальном времени для предварительно амплифицированного фрагмента гена антибиотикоустойчивости bla-NDM-1, обработанного рибонуклеопортеиновым комплексом, содержащим направляющую РНК crRNA blaNDM №760 и белок LbCpf1, на котором посредством графика визуализировано выявление фрагмента модельной матрицы pGEM-T-bla-NDM-1 с помощью рибонуклеопротеиновых комплексов LbCpf1 из Lachnospiraceae, содержащих направляющую РНК crRNA blaNDM №760, где:
- по вертикали указаны значения нормализированного флуоресцентного сигнала, создаваемого красителем;
- по горизонтали указано время флуоресценции, в минутах;
- на кривых 65-72 показано количество копий нуклеиновой кислоты на реакцию, а именно:
65 - 3480000 копий/реакция;
66 - 348000 копий/реакция;
67 - 34800 копий/реакция;
68 - 3480 копий/реакция;
69 - 348 копий/реакция;
70 - 34,8 копий/реакция;
71 - 3,48 копий/реакция;
72 - К- (отрицательный контроль).
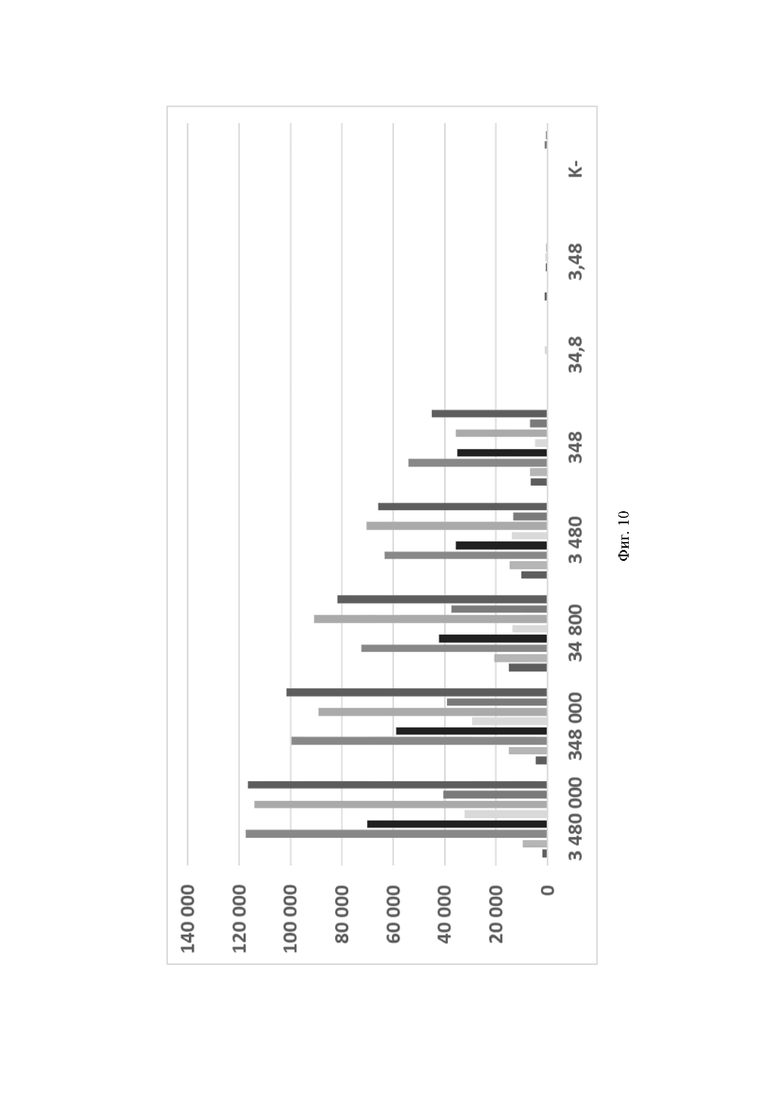
Фиг. 10. Значения флуоресценции в конечной точке (60 цикл анализа, 60 минут) для предварительно амплифицированного фрагмента гена антибиотикоустойчивости bla-NDM-1, обработанного рибонуклеопортеиновыми комплексами, содержащим направляющие РНК crRNA blaNDM №100, crRNA blaNDM №124, crRNA blaNDM №204, crRNA blaNDM №285, crRNA blaNDM №540, crRNA blaNDM №583, crRNA blaNDM №666 и crRNA blaNDM №760, где посредством графика визуализировано выявление фрагмента модельной матрицы pGEM-T-bla-NDM-1 с помощью рибонуклеопротеиновых комплексов LbCpf1 из Lachnospiraceae, при этом:
- по вертикали указаны значения нормализированного флуоресцентного сигнала, создаваемого красителем;
- по горизонтали указано количество копий модельной матрицы в реакции;
- столбцом  визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №100;
визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №100;
- столбцом визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №124;
- столбцом  визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №204;
визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №204;
- столбцом  визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №285;
визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №285;
- столбцом визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №540;
- столбцом визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №583;
- столбцом  визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №666;
визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №666;
- столбцом  визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №760.
визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №760.
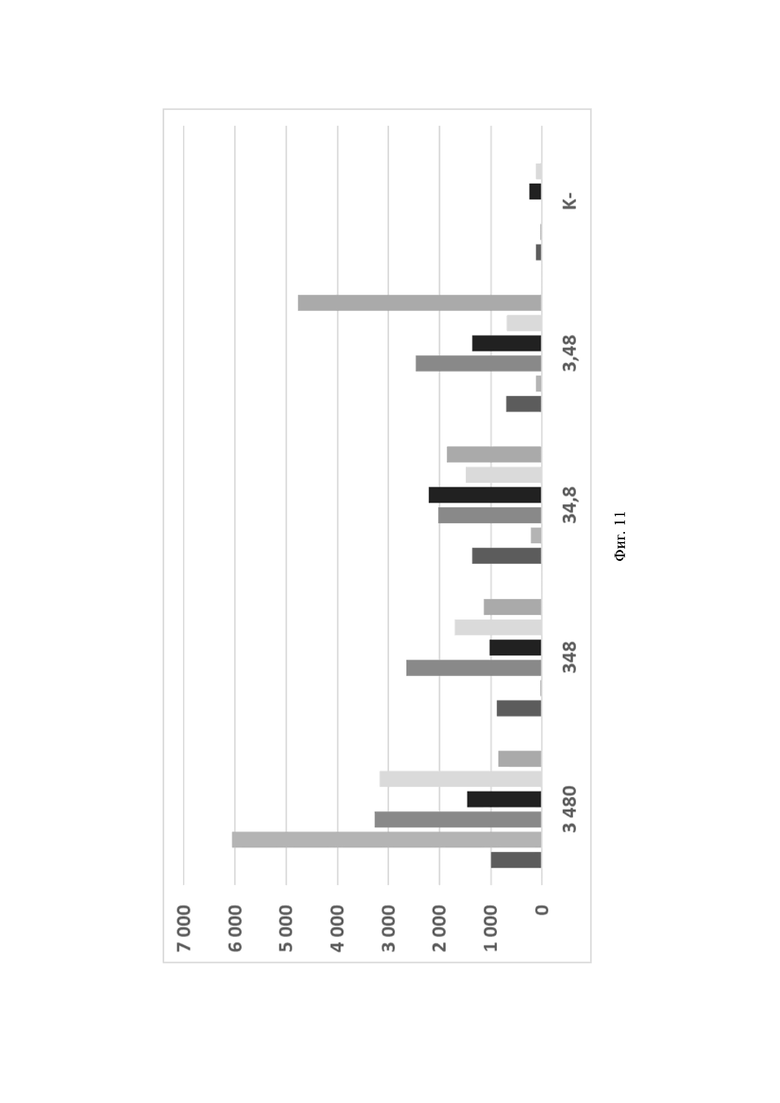
Фиг. 11. Значения флуоресценции в конечной точке (60 цикл анализа, 60 минут) для предварительно амплифицированного фрагмента гена антибиотикоустойчивости bla-NDM-1, обработанного рибонуклеопортеиновыми комплексами, содержащим направляющие РНК crRNA blaNDM №124, crRNA blaNDM №204, crRNA blaNDM №285, crRNA blaNDM №583, crRNA blaNDM №666 и crRNA blaNDM №760, где посредством графика визуализировано выявление фрагмента модельной матрицы pGEM-T-bla-NDM-1 с помощью рибонуклеопротеиновых комплексов LbCpf1 из Lachnospiraceae, при этом:
- по вертикали указаны значения нормализированного флуоресцентного сигнала, создаваемого красителем;
- по горизонтали указано количество копий модельной матрицы в реакции;
- столбцом  визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №124;
визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №124;
- столбцом визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №204;
- столбцом I визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №285;
- столбцом I визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №583;
- столбцом визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №666;
- столбцом визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №760.
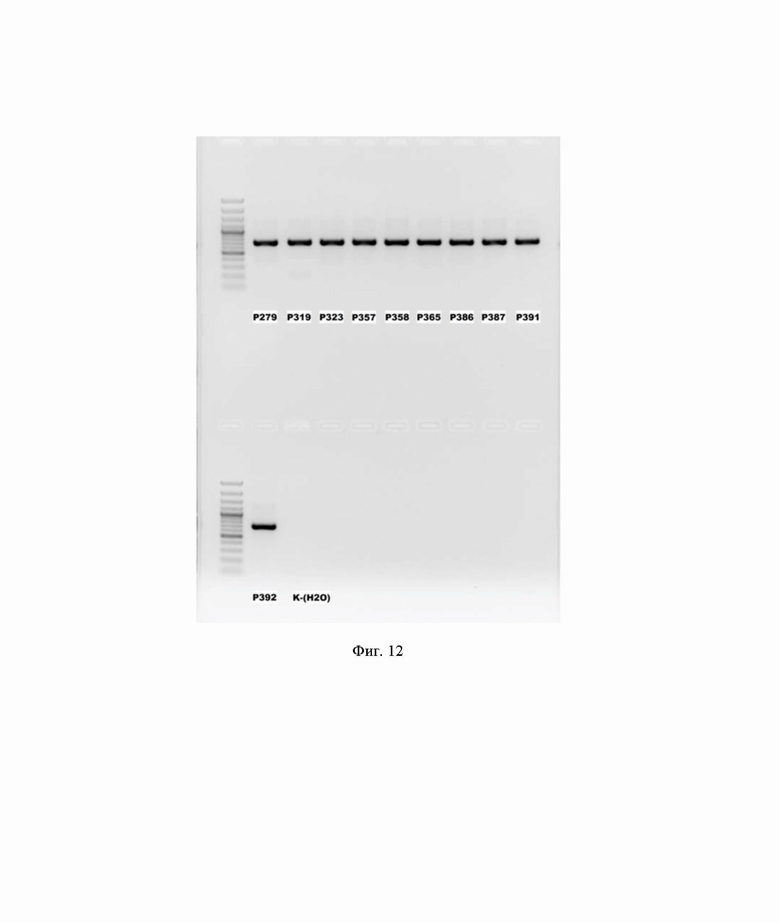
Фиг. 12. Визуализация амплифицированного с клинических образцов фрагмента гена антибиотикоустойчивости bla-NDM-1 (размером 680 п.о.) после предварительной амплификации с использованием олигонуклеотидов NDM-1 for 1 и NDM-1 rev 1 при помощи электрофореза в агарозном геле.
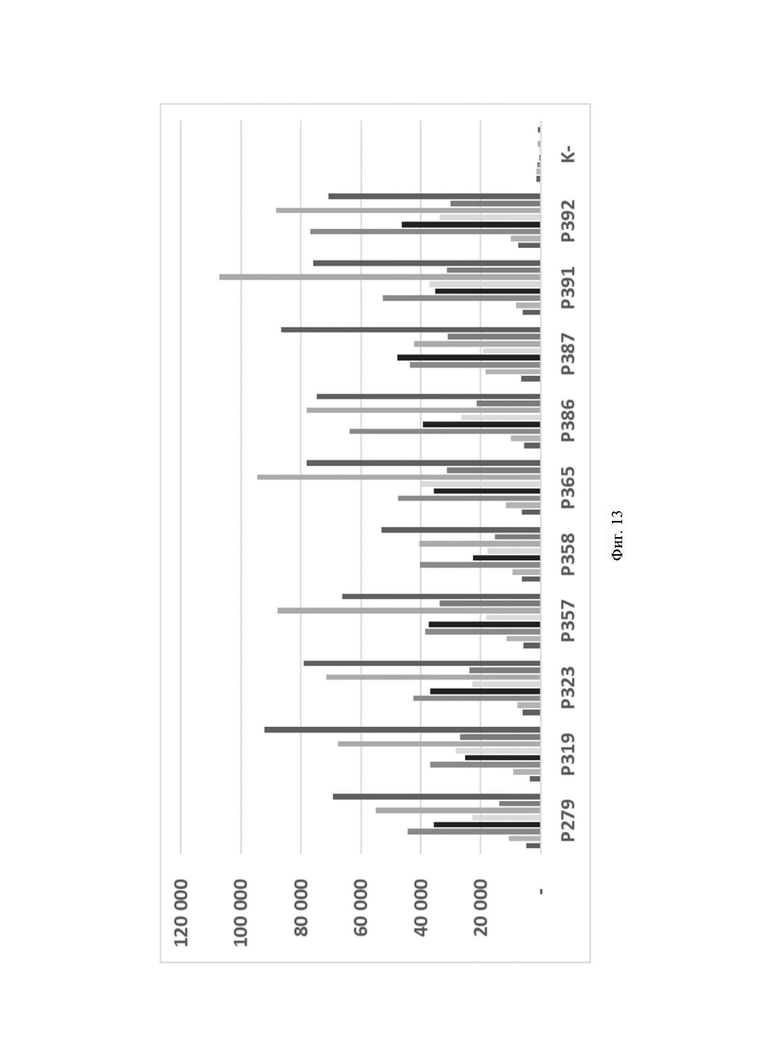
Фиг. 13. Значения флуоресценции в конечной точке (60 цикл анализа, 60 минут) для предварительно амплифицированного с клинических образцов фрагмента гена антибиотикоустойчивости bla-NDM-1, обработанного рибонуклеопортеиновыми комплексами, содержащим направляющие РНК crRNA blaNDM №100, crRNA blaNDM №124, crRNA blaNDM №204, crRNA blaNDM №285, crRNA blaNDM №540, crRNA blaNDM №583, crRNA blaNDM №666 и crRNA blaNDM №760, где посредством графика визуализировано выявление фрагмента гена антибиотикоустойчивости bla-NDM-1 с помощью рибонуклеопротеиновых комплексов LbCpf1 из Lachnospiraceae на ограниченной панели клинических образцов, при этом:
- по вертикали указаны значения нормализированного флуоресцентного сигнала, создаваемого красителем;
- по горизонтали указаны идентификаторы образца;
- столбцом  визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №100;
визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №100;
- столбцом визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №124;
- столбцом визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №204;
визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №204;
- столбцом  визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №285;
визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №285;
- столбцом визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №540;
- столбцом визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №583;
- столбцом  визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №666;
визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №666;
- столбцом  визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №760.
визуализирована эффективность выявления единичных копий фрагмента модельной матрицы pGEM-T-bla-NDM-1 с использованием crRNA blaNDM №760.
Примеры осуществления изобретения
ПРИМЕР 1: ПОДБОР ПОСЛЕДОВАТЕЛЬНОСТЕЙ-МИШЕНЕЙ В ГЕНЕ АНТИБИОТИКОУСТОЙЧИВОСТИ BLA-NDM-1 ДЛЯ СОЗДАНИЯ НАПРАВЛЯЮЩИХ РНК
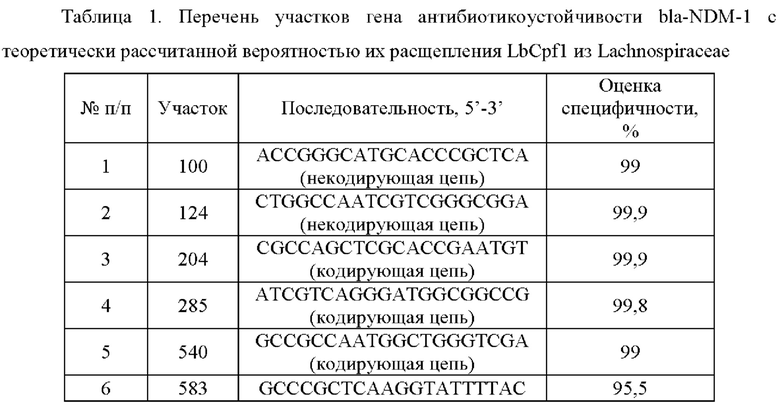
Для подбора последовательностей-мишеней в гене антибиотикоустойчивости blaNDM-1 для создания направляющих РНК были использованы современные алгоритмы in silico анализа нуклеотидных последовательностей и программы, находящиеся в открытом доступе, включая Benchling (https://www.benchling.com/molecular-biology/). Был составлен перечень участков гена антибиотикоустойчивости bla-NDM-1 с теоретически рассчитанной вероятностью их расщепления в высоко консервативных участках (Таблица 1). Направляющие РНК, специфически узнающие высоко консервативные участки гена антибиотикоустойчивости bla-NDM-1, представлены уникальными последовательностями SEQ ID NO: 1-8.

ПРИМЕР 2: ПОДГОТОВКА МАТЕРИАЛА ДЛЯ ОБНАРУЖЕНИЯ ГЕНА АНТИБИОТИКОУСТОЙЧИВОСТИ BLA-NDM-1 МЕТОДОМ ПРЕДВАРИТЕЛЬНОЙ АМПЛИФИКАЦИИ
Подготовку материала для обнаружения гена антибиотикоустойчивости bla-NDM-1 проводили методом предварительной амплификации. В качестве модельной матрицы, содержащей фрагмент гена антибиотикоустойчивости bla-NDM-1, использовали плазмидную ДНК pGEM-T-bla-NDM-1, содержащую в своем составе фрагмент гена антибиотикоустойчивости антибиотикоустойчивости bla-NDM-1 размером 720 п.о.
Предварительную амплификацию участка, соответствующего фрагменту гена антибиотикоустойчивости bla-NDM-1, проводили с использованием специфических олигонуклеотидов с SEQ ID NO: 9 и SEQ ID NO: 10.
Продукт, кодирующий фрагмент гена антибиотикоустойчивости bla-NDM-1, получали при проведении ПЦР с использованием специфических олигонуклеотидов NDM-1_for_1 и NDM-1_rev_1 (ГенТерра, Россия) и полимеразы TaqF (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия). Размер амплифицированного фрагмента составлял 680 пар нуклеотидов.
Температурный профиль амплификации для получения ПЦР-продуктов фрагментов гена антибиотикоустойчивости bla-NDM-1:
1. денатурация: 95°С в течение 3 мин;
2. 40 циклов амплификации: 95°С - 15 с, 55°С - 45 с, 72°С - 30 с;
3. финальная элонгация: 72°С в течение 5 мин.
В ходе подготовки материала для обнаружения гена антибиотикоустойчивости blaNDM-1 методом предварительной амплификации проводили титрование модельной матрицы pGEM-T-bla-NDM-1 путем приготовления серийных разведений (Таблица 2).

Для оценки эффективности предварительной амплификации полученный фрагмент гена антибиотикоустойчивости bla-NDM-1 визуализировали при помощи электрофореза в агарозном геле (фиг.1).
Подготовленный описанным способом материал использовали для экспериментов по выявлению гена антибиотикоустойчивости bla-NDM-1 с помощью рибонуклеопротеиновых комплексов LbCpf1 из Lachnospiraceae, содержащих направляющие РНК crRNA blaNDM №100, crRNA blaNDM №124, crRNA blaNDM №204, crRNA blaNDM №285, crRNA blaNDM №540, crRNA blaNDM №583, crRNA blaNDM №666 и crRNA blaNDM №760, без предварительной очистки.
ПРИМЕР 3: СОЗДАНИЕ РИБОНУКЛЕОПРОТЕИНОВЫХ КОМПЛЕКСОВ ДЛЯ ОБНАРУЖЕНИЯ ГЕНА АНТИБИОТИКОУСТОЙЧИВОСТИ BLA-NDM-1
В качестве направляющих РНК для обнаружения гена антибиотикоустойчивости bla-NDM-1 были использованы специфические РНК-олигонуклеотиды, приведенные в Таблице 3.

Создание готового рибонуклеопротеинового комплекса, содержащего белок семейства CRISPR-Cas 12 LbCpf1 из Lachnospiraceae, и направляющую РНК авторы проводили по стандартному протоколу с некоторыми модификациями [С. Anders, М. Jinek, In vitro enzymology of Cas9, Methods Enzymol. 546 (2014) 1-20, https://doi.org/10.1016/B978-0-12-801185-0.00001-5]. Для формирования готового рибонуклеопротеинового комплекса 250 нг Cas-белка LbCpf1 из Lachnospiraceae и направляющую РНК смешивали и инкубировали 10 минут при комнатной температуре. Полученный таким способом рибонуклеопротеиновый комплекс готов для выявления гена антибиотикоустойчивости bla-NDM-1.
ПРИМЕР 4: ОБНАРУЖЕНИЕ ЕДИНИЧНЫХ КОПИЙ ГЕНА АНТИБИОТИКОУСТОЙЧИВОСТИ BLA-NDM-1 С ПОМОЩЬЮ РИБОНУКЛЕОПРОТЕИНОВЫХ КОМПЛЕКСОВ CRTSPR7CAS НА ПРИМЕРЕ МОДЕЛЬНОЙ МАТРИЦЫ
Предварительно амплифицированный материал, полученный способом, описанным в Примере 2, использовали в качестве матрицы для обнаружения гена антибиотикоустойчивости bla-NDM-1 с помощью рибонуклеопротеиновых комплексов CRISPR-Cas, полученных способом, описанным в Примере 3.
Для обнаружения гена антибиотикоустойчивости bla-NDM-1 с помощью рибонуклеопротеиновых комплексов CRISPR-Cas готовили реакционную смесь, содержащую следующие компоненты:
10 х буфер (100 mM TrisHCl рН 8,0, 1 М NaCl);
50 mM MgCl2 (конечная концентрация в реакционной смеси 10 mM);
250 нг рибонуклеопротеинового комплекса (LbCpf1 из Lachnospiraceae и направляющие РНК crRNA blaNDM №100, crRNA blaNDM №124, crRNA blaNDM №204, crRNA blaNDM №285, crRNA blaNDM №540, crRNA blaNDM №583, crRNA blaNDM №666 и crRNA blaNDM №760);
10 pmol флуоресцентный зонд (6FAM-TTATT-BHQ1);
Мишень (предварительно амплифицированный фрагмент гена антибиотикоустойчивости bla-NDM-1);
вода mQ.
Реакционные смеси, содержащие все необходимые компоненты, помещали в амплификатор QuantStudio 5 (Thermo Fisher Scientific, США) и задавали следующие параметры реакции:
30-60 циклов:
1. 37°С-35 сек,
2. 37°С - 25 сек, съемка флуоресценции.
В первую очередь были проведены эксперименты по обнаружению единичных копий гена антибиотикоустойчивости bla-NDM-1 с помощью рибонуклеопротеиновых комплексов CRISPR-Cas, сформированных на основе LbCpf1 из Lachnospiraceae, с использованием в качестве мишени модельной матрицы плазмидной ДНК pGEM-T-bla-NDM-1, содержащую в своем составе фрагмент гена антибиотикоустойчивости bla-NDM-1 размером 720 п.о. На Фиг. 2-9 приведены типичные результаты анализа на примерах профилей флуоресценции в реальном времени для предварительно амплифицированного фрагмента гена антибиотикоустойчивости bla-NDM-1, обработанного рибонуклеопортеиновыми комплексами, содержащими направляющие РНК crRNA blaNDM №100, crRNA blaNDM №124, crRNA blaNDM №204, crRNA blaNDM №285, crRNA blaNDM №540, crRNA blaNDM №583, crRNA blaNDM №666 и crRNA blaNDM №760 и белок LbCpf1. Наилучший результат выявление 348 копий модельной матрицы в реакционной смеси был показан для всех использованных направляющих РНК.
Для обнаружения единичных копий гена антибиотикоустойчивости bla-NDM-1 с помощью рибонуклеопротеиновых комплексов CRISPR-Cas было принято решение оптимизировать процесс предварительной амплификации. Для этого были разработаны дополнительные олигонуклеотиды с SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 13 и SEQ ID NO: 14 для амплификации фрагмента гена антибиотикоустойчивости bla-NDM-1, входящего в состав модельной матрицы pGEM-T-bla-NDM-1. Полученные с помощью оптимизированных условий продукты амплификации использовали в качестве матрицы для обнаружения единичных копий гена антибиотикоустойчивости bla-NDM-1 с помощью рибонуклеопротеиновых комплексов CRISPR-Cas, полученных способом, описанным в Примере 3.
В ходе проведенного анализа было показано, что рибонуклеопротеиновые комплексы CRISPR-Cas, сформированные на основе LbCpf1 из Lachnospiraceae и направляющих РНК crRNA blaNDM №124, crRNA blaNDM №204, crRNA blaNDM №285, crRNA blaNDM №583, crRNA blaNDM №666 и crRNA blaNDM №760 обладают способностью выявлять единичные копии гена антибиотикоустойчивости bla-NDM-1 (3,48 копии/реакция). Применение оптимизированных олигонуклеотидов позволило обнаруживать до 3,48 копий ДНК, внесенных в реакцию предварительной амплификации (фиг.11). При этом в среднем уже на 25 цикле (25 минут) анализа значение сигнала превышало значение «шума» (неспецифической флуоресценции контрольного образца, не содержащего мишени) в 5 раз, а к 35 циклу - более чем в 10 раз.
Таким образом, разработанные направляющие РНК позволяют ультрачувствительно выявлять единичные копии гена антибиотикоустойчивости bla-NDM-1 в образце после предварительной амплификации в составе рибонуклеопротеиновых комплексов CRISPR-Cas.
В ходе работ была оценена эффективность выявления гена антибиотикоустойчивости bla-NDM-1, содержащегося в составе модельной матрицы, с использованием различных направляющих РНК. Было показано, что рибонуклеопротеиновые комплексы CRISPR-Cas, сформированные на основе LbCpf1 из Lachnospiraceae и направляющих РНК, выявляют ген антибиотикоустойчивости bla-NDM-1 с различной эффективностью, и их можно расположить в следующем порядке по уменьшению активности: crRNA blaNDM №760>crRNA blaNDM №285>crRNA blaNDM №583>crRNA blaNDM №666>crRNA blaNDM №124>crRNA blaNDM №204 (Фиг. 11).
ПРИМЕР 5: ОБНАРУЖЕНИЕ ГЕНА АНТИБИОТИКОУСТОЙЧИВОСТИ BLANDM-1 С ПОМОЩЬЮ РИБОНУКЛЕОПРОТЕИНОВЫХ КОМПЛЕКСОВ CRISPR/CAS НА ОГРАНИЧЕННОЙ ПАНЕЛИ КЛИНИЧЕСКИХ ОБРАЗЦОВ
Разработанные направляющие РНК были апробированы на ограниченной панели клинических образцов (10 шт.), содержащих ген антибиотикоустойчивости bla-NDM-1 (ранее подтверждено методом секвенирования следующего поколения).
Для обнаружения гена антибиотикоустойчивости bla-NDM-1 с помощью рибонуклеопротеиновых комплексов CRISPR-Cas была проведена предварительная амплификация фрагмента гена антибиотикоустойчивости bla-NDM-1. Для проведения предварительной амплификации из клинических образцов 10 пациентов с помощью коммерчески доступного набора «РИБО-преп» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) согласно инструкции производителя были выделены препараты ДНК.
Продукт, кодирующий фрагмент гена антибиотикоустойчивости bla-NDM-1, получали с помощью ПЦР с использованием специфических олигонуклеотидов NDM-1_for_1 и NDM-1_rev_1 (ГенТерра, Россия) и визуализировали при помощи электрофореза в агарозном геле (Фиг. 12).
Полученный таким способом материал использовали в качестве матрицы для обнаружения гена антибиотикоустойчивости bla-NDM-1 с помощью рибонуклеопротеиновых комплексов CRISPR-Cas, полученных способом, описанным в Примере 3.
Обнаружение гена антибиотикоустойчивости bla-NDM-1 с помощью рибонуклеопротеиновых комплексов CRISPR-Cas проводили способом, описанным в Примере 4.
В ходе проведенного анализа было показано, что рибонуклеопротеиновые комплексы CRISPR-Cas обладают способностью выявлять ген антибиотикоустойчивости bla-NDM-1 в препаратах ДНК, выделенных из клинических образцов. При этом в среднем уже на 10 цикле (10 минут) анализа значение сигнала превышало значение «шума» (неспецифической флуоресценции контрольного образца, не содержащего мишени) более чем в 5 раз, а к 16 циклу (16 минут) анализа - более чем в 10 раз (Таблица 4).
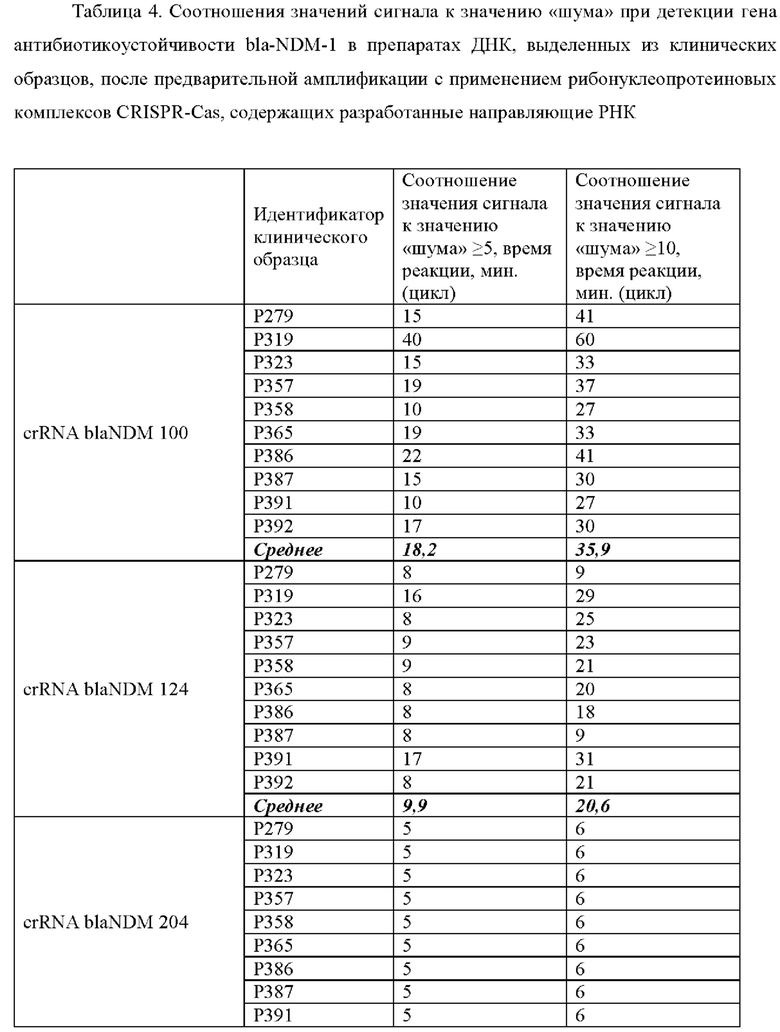
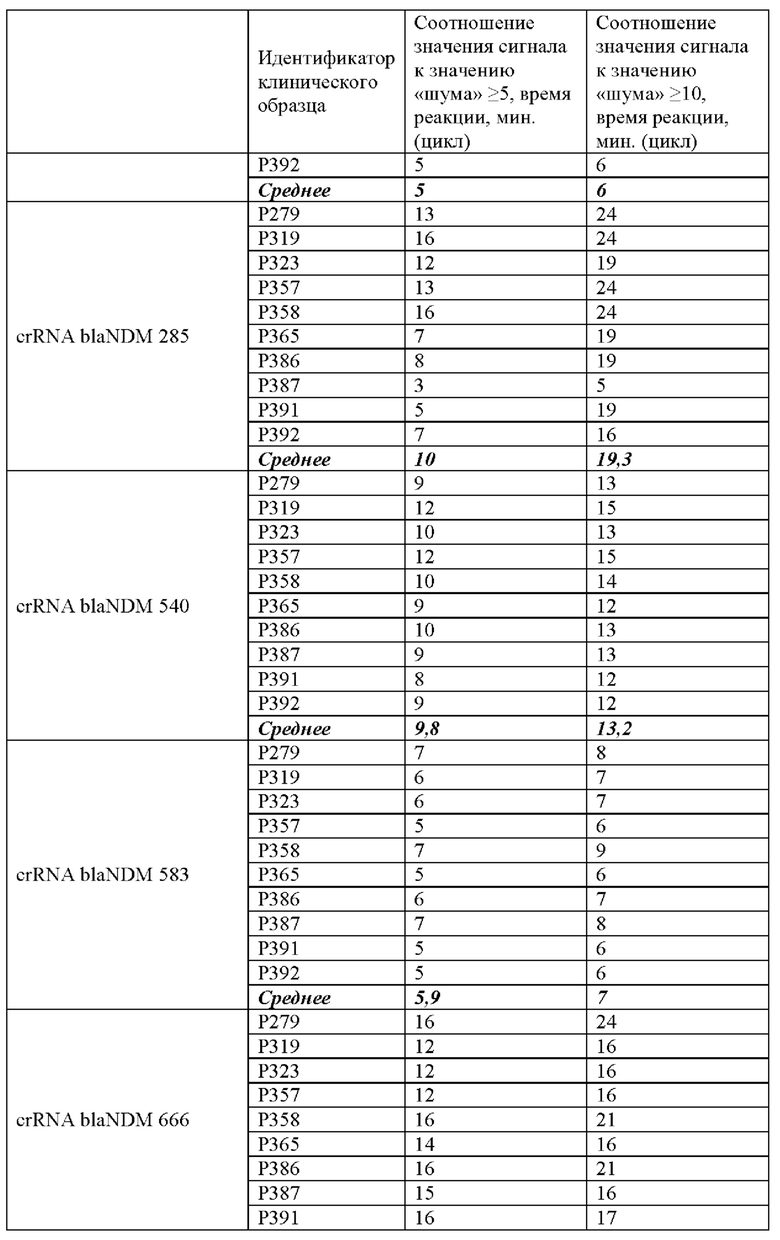
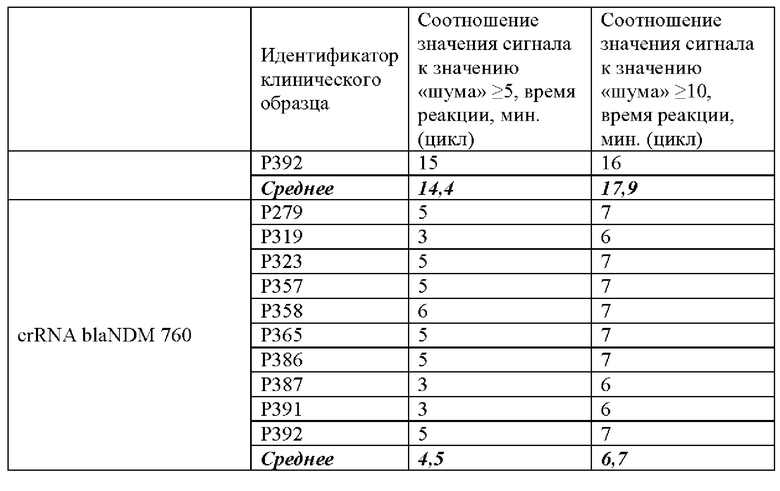
Типичные результаты анализа приведены на примерах значений флуоресценции в конечной точке (60 цикл анализа, 60 минут) для предварительно амплифицированного фрагмента гена антибиотикоустойчивости bla-NDM-1 (10 независимых клинических образцов), обработанных рибонуклеопортеиновыми комплексами, содержащими направляющие РНК crRNA blaNDM №100, crRNA blaNDM №124, crRNA blaNDM №204, crRNA blaNDM №285, crRNA blaNDM №540, crRNA blaNDM №583, crRNA blaNDM №666 и crRNA blaNDM №760 и белок LbCpf1, на Фиг. 13.
Эффективность выявления гена антибиотикоустойчивости bla-NDM-1, содержащегося в составе препаратов ДНК, выделенных из клинических образцов, с использованием различных направляющих РНК в составе рибонуклеопротеиновых комплексов CRISPR-Cas, сформированных на основе LbCpf1 из Lachnospiraceae, оцененная по соотношению значений сигнала к значению «шума» при детекции, можно представить в следующем порядке по убыванию: crRNA blaNDM №760≥crRNA blaNDM №204≥crRNA blaNDM №583>crRNA blaNDM №540≥crRNA blaNDM №124≥crRNA blaNDM №285>crRNA blaNDM №666>crRNA blaNDM №100 (Таблица 4).
Таким образом, разработанные направляющие РНК позволяют ультрачувствительно выявлять единичные копии гена антибиотикоустойчивости bla-NDM-1, и способны обнаруживать его препаратах ДНК, выделенных из клинических образцов, после предварительной амплификации в составе рибонуклеопротеиновых комплексов CRISPR-Cas.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.2//EN" "ST26SequenceListing_V1_2.dtd">
<ST26SequenceListing dtdVersion="V1_2" fileName="Sposob polucheniya
preparata ribonukleoproteinovogo kompleksa CRISPR-Cas i preparat dlya
vyyavleniya gena antibiotikoustojchivosti bla-NDM-1 v
ul'tranizkih koncentraciyah" softwareName="WIPO Sequence"
softwareVersion="1.0.0" productionDate="2024-09-26">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>nn</ApplicationNumberText>
<FilingDate>2024-09-25</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>nn</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>nn</ApplicationNumberText>
<FilingDate>2024-09-25</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">ФБУН ЦНИИ Эпидемиологии
Роспотребнадзора</ApplicantName>
<ApplicantNameLatin>FBUN CRIE</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ получения препарата
рибонуклеопротеинового комплекса CRISPR-Cas и препарат для выявления
гена антибиотикоустойчивости bla-NDM-1 в ультранизких
концентрациях</InventionTitle>
<SequenceTotalQuantity>14</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>blaNDM</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aatttctactaagtgtagataccgggcatgcacccgctca</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>blaNDM</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aatttctactaagtgtagatctggccaatcgtcgggcgga</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>blaNDM</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aatttctactaagtgtagatcgccagctcgcaccgaatgt</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>blaNDM</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aatttctactaagtgtagatatcgtcagggatggcggccg</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>blaNDM</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aatttctactaagtgtagatgccgccaatggctgggtcga</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>blaNDM</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aatttctactaagtgtagatgcccgctcaaggtattttac</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>blaNDM</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aatttctactaagtgtagatggtggctgcctgatcaagga</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>blaNDM</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aatttctactaagtgtagatgtgcggcgttccccaaggcc</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>NDM-1</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctgcattgatgctgagcgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>NDM-1</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgctggccttggggaacgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>32</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>NDM-1</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agctgagcaccgcattagccgctgcattgatg</INSDSeq_sequenc
e>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>34</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>NDM-1</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcgcgctggcggtggtgactcacgcgcatcagg</INSDSeq_seque
nce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>36</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>NDM-1</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctggttcgacaacgcattggcataagtcgcaatcc</INSDSeq_seq
uence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>33</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..33</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>NDM-1</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cggaatggctcatcacgatcatgctggccttgg</INSDSeq_sequen
ce>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Система CRISPR-Cas12 для выявления гена антибиотикоустойчивости bla-NDM-1 в ультранизких концентрациях | 2024 |
|
RU2839761C1 |
| Способ обнаружения гена антибиотикоустойчивости bla-NDM-1 в ультранизких концентрациях и специфические олигонуклеотиды для использования в способе | 2024 |
|
RU2841369C1 |
| Набор CRISPR-Cas12 для выявления гена антибиотикоустойчивости bla-NDM-1 в ультранизких концентрациях (варианты) | 2024 |
|
RU2839482C1 |
| Способ получения препарата рибонуклеопротеинового комплекса CRISPR-Cas и препарат для выявления гена антибиотикоустойчивости bla-TEM-1B в ультранизких концентрациях | 2023 |
|
RU2820307C1 |
| Способ получения препарата рибонуклеопротеинового комплекса CRISPR-Cas и препарат для выявления гена антибиотикоустойчивости bla-OXA-1 в ультранизких концентрациях | 2024 |
|
RU2839763C1 |
| Система CRISPR-Cas12 для выявления гена антибиотикоустойчивости bla-TEM-1B в ультранизких концентрациях | 2023 |
|
RU2829103C1 |
| Система CRISPR-Cas12 для выявления гена антибиотикоустойчивости bla-OXA-1 в ультранизких концентрациях | 2024 |
|
RU2839762C1 |
| Набор CRISPR-Cas12 для выявления гена антибиотикоустойчивости bla-OXA-1 в ультранизких концентрациях (варианты) | 2024 |
|
RU2839486C1 |
| Способ обнаружения гена антибиотикоустойчивости bla-OXA-1 в ультранизких концентрациях и специфические олигонуклеотиды для использования в способе | 2024 |
|
RU2841370C1 |
| Способ обнаружения гена антибиотикоустойчивости bla-TEM-1B в ультранизких концентрациях и специфические олигонуклеотиды для использования в способе | 2023 |
|
RU2820306C1 |
Изобретение относится к области биотехнологии, в частности к cпособу получения препарата рибонуклеопротеинового комплекса CRISPR-Cas. Указанный способ содержит стадии: (i) объединения Cas-белка семейства CRISPR-Cas LbCpf1 из Lachnospiraceae в комплекс с по меньшей мере одной направляющей РНК с SEQ ID NO: 1-8; и при необходимости (ii) лиофильной сушки указанного комплекса. Кроме того, изобретение также относится к препарату рибонуклеопротеинового комплекса CRISPR-Cas для выявления гена антибиотикоустойчивости bla-NDM-1, полученному указанным способом. Настоящее изобретение обеспечивает значительное повышение эффективности выявления гена антибиотикоустойчивости bla-NDM-1. 2 н. и 2 з.п. ф-лы, 13 ил., 4 табл., 5 пр.
1. Способ получения препарата рибонуклеопротеинового комплекса CRISPR-Cas, отличающийся тем, что содержит стадии:
(i) объединения Cas-белка семейства CRISPR-Cas LbCpf1 из Lachnospiraceae в комплекс с по меньшей мере одной направляющей РНК с SEQ ID NO: 1-8; и при необходимости
(ii) лиофильной сушки рибонуклеопротеинового комплекса CRISPR-Cas, полученного на стадии (i);
с получением, таким образом, препарата рибонуклеопротеинового комплекса CRISPR-Cas.
2. Препарат рибонуклеопротеинового комплекса CRISPR-Cas для выявления гена антибиотикоустойчивости bla-NDM-1, полученный способом по п.1, содержащий Cas-белок семейства CRISPR-Cas LbCpf1 из Lachnospiraceae в комплексе с по меньшей мере одной направляющей РНК с SEQ ID NO: 1-8.
3. Препарат по п. 2, где препарат представляет собой раствор указанного рибонуклеопротеинового комплекса CRISPR-Cas.
4. Препарат по п. 2, где указанный препарат лиофилизирован.
| М.А.ТЮМЕНЦЕВА и др., CRISPR/Cas-белки для выявления генов антибиотикоустойчивости у патогенных микроорганизмов, Бактериология, 2023, т | |||
| Топка с несколькими решетками для твердого топлива | 1918 |
|
SU8A1 |
| Способ очищения сернокислого глинозема от железа | 1920 |
|
SU47A1 |
| Способ получения препарата рибонуклеопротеинового комплекса CRISPR/Cas и препарат для выявления гена антибиотикоустойчивости bla VIM-2 (металло-бета-лактамаза класс B VIM-2) Pseudomonas aeruginosa в ультранизких концентрациях | 2020 |
|
RU2745637C1 |
| CN 110923230 A, 27.03.2020 | |||
| THAPA, A | |||
| et al., Detection of NDM Variants (blaNDM-1, blaNDM-2, blaNDM-3) from Carbapenem-Resistant Escherichia coli and Klebsiella | |||
Авторы
Даты
2025-05-05—Публикация
2024-09-30—Подача