Область техники
Изобретение относится к области биотехнологии, молекулярной биологии и генной инженерии, а именно, к способу идентификации ДНК возбудителей острых кишечных инфекций (ОКИ), включающей этапы выделения и амплификации ДНК, последующей гибридизации амплифицированной ДНК на биологическом микрочипе. Изобретение также раскрывает состав высокоспецифичных ДНК-зондов и олигонуклеотидов для ПЦР, набор реактивов для идентификации.
Уровень техники
Выявление и идентификация ДНК возбудителей инфекционных заболеваний желудочно-кишечного тракта представляет актуальную проблему Острые кишечные инфекции (ОКИ) продолжают занимать существенное место в структуре инфекционных заболеваний, уступая по распространенности лишь острым респираторным вирусным инфекциям. Ежегодно в мире число заболевших ОКИ достигает в 3-5 млрд. человек, при этом 5-10 млн. больных умирают. Как в развивающихся, так и в индустриально развитых странах, несмотря на определенные успехи в диагностике и лечении, проблема ОКИ стоит достаточно остро.
В настоящее время более перспективным для диагностики патогенных бактерий является применение метода ПЦР и применение ДНК-микрочипов [1, 2].
Известен патент США №7,364,898 в котором рассматривается возможность идентификации с помощью микрочипа бактерий входящих в группу состоящую из Staphylococcus, Enterococcus, Streptococcus, Haemolyticus, Pseudomonas, Campylobacter, Enterobacter, Neisseria, Proteus, Salmonella, Simonsiella, Riemerella, Escherichia, Neisseria, Meningococcus, Moraxella, Kingella, Chromobacterium, Branhamella [3].
Известна заявка на изобретение в США №20060240453, в которой заявляется о возможности идентификации широкой группы бактерий, включая Escheria coli, Klebsiella pneumoniae, Salmonella, Campylobacter, Shigella species, enteroinvasive E. Coli [4].
Известна заявка США №20060194206, в которой описан способ идентификации диареи вызываемой Shigella и патогенной E.coli (DEC) с помощью нескольких специфических генетических маркеров, которые входят в группу, состоящую из А/ЕЕС & ЕРЕС, ETEC, VTEC, EIEC, ehxA, ipaH генов. Согласно изобретению проводят мультипраймерную ПЦР. Продукт ПЦР может детектироваться или с помощью капиллярного электрофореза, либо на поверхности мембран (ELISA) либо ДНК чипов, или с помощью Luminex.RTM [5].
Наиболее близкой к рассматриваемой диагностике является заявка на патент RU №2004114259 [6], в которой рассматривается обнаружение и идентификация кишечных бактерий с помощью микрочипа.
Одним из недостатков рассматриваемых микрочипов и способов идентификации является низкий уровень контроля за процессом диагностики или его отсутствие и трудности в реализации способа идентификации из-за слабой отработки отдельных стадий, что требует дальнейших усилий. Например, к недостатку изобретения RU №2004114259 можно отнести тот факт, что в перечне маркеров нет маркеров положительного и отрицательного контролей.
Кроме того, обычно в данных работах используется либо двухшаговая ПЦР, либо используется ассиметричная мультипраймерная ПЦР, которая достаточно затратна. Известен способ идентификации на основе биочипа, в котором используют более простую симметричную мультипраймерную ПЦР и контроль за прохождением процесса диагностики, но данный чмп разработан для идентификации ДНК трансгенных растений [7].
Техническая задача изобретения состоит в том, чтобы создать более совершенную диагностику ОКИ с использованием нового биологического микрочипа, обладающего более совершенными характеристиками при невысокой стоимости скрининга наличия ОКИ.
Другая задача состоит в увеличении достоверности и воспроизводимости получаемых результатов. Эта задача решается за счет применения в биологических чипах для диагностики ОКИ новых, более эффективных последовательностей праймеров и за счет разработки способа обработки продуктов мультипраймерной симметричной ПЦР ферментом, повышающим эффективность гибридизации, а также за счет размещения кластеров положительного контроля одновременно выполняющих роль идентификаторов границ рабочей зоны биочипа.
Следующей задачей является разработка набора для проведения диагностики, в состав которого входят новые праймеры, новый биочип и фермент для обработки продуктов ПЦР.
Сущность изобретения
Предлагаемый метод диагностики основан на проведении мультипраймерной симметричной ПЦР с использованием специфических праймеров, комплементарных последовательностям генов-маркеров ОКИ, и последующей гибридизации продуктов ПЦР на микрочипе, содержащем оригинальный набор дифференцирующих олигонуклеотидов.
Одним из объектов изобретения является способ идентификации маркеров острых кишечных инфекций, включающий получение образца исследуемой ДНК, прямых и обратных праймеров, специфичных к ДНК одного или более маркеров ОКИ, и микрочипа, проведение ПЦР в присутствии образца ДНК и праймеров, специфичных к указанному одному или более маркерам, нанесение ПЦР-продуктов на рабочую зону биочипа, инкубацию в условиях гибридизации, детекцию образованных гибридизационных комплексов на биочипе. В способе используют микрочип, который содержит дифференцирующие олигонуклеотиды, последовательности которых представлены в SEQ ID NO: 3, 6, 9, 12, 15, 18, 19, приведенных в таблице 3 и перечне последовательностей, а в качестве специфических праймеров используют олигонуклеотиды, последовательность которых выбирают из группы, состоящей из SEQ ID NO: 1, 2, 4, 5, 7, 8, 10, 11, 13, 14, 16, 17 приведенных в таблице 2 и перечне последовательностей, проводят мультипраймерную ПЦР, причем в паре прямого и обратного праймеров один содержит метку, которую после гибридизации ДНК детектируют, регистрируют и используют для идентификации маркеров ОКИ.
В части выбора праймеров для ПЦР предложен набор из 5 пар олигонуклеотидов, комплементарных нуклеотидным последовательностям генов-маркеров, включенных в структуру патогенных микроорганизмов, относящихся к группе родов Shigella spp. и Enferoinvasive E.coli (EIEC), Salmonella spp., Campylobacter jejuni, Proteus mirabilis и Klebsiella pneumoniae. Выбранные участки маркеров приведены в таблице 1, а последовательности специфических олигонуклеотидов, используемых в качестве праймеров для выявления маркеров в способе идентификации ОКИ, представлены в таблице 2.
Другим объектом изобретения являются дифференцирующие олигонуклеотиды, последовательность которых представлена в таблице 3 и используемых в качестве зондов, иммобилизованных на поверхности биочипа.
Перечень фигур
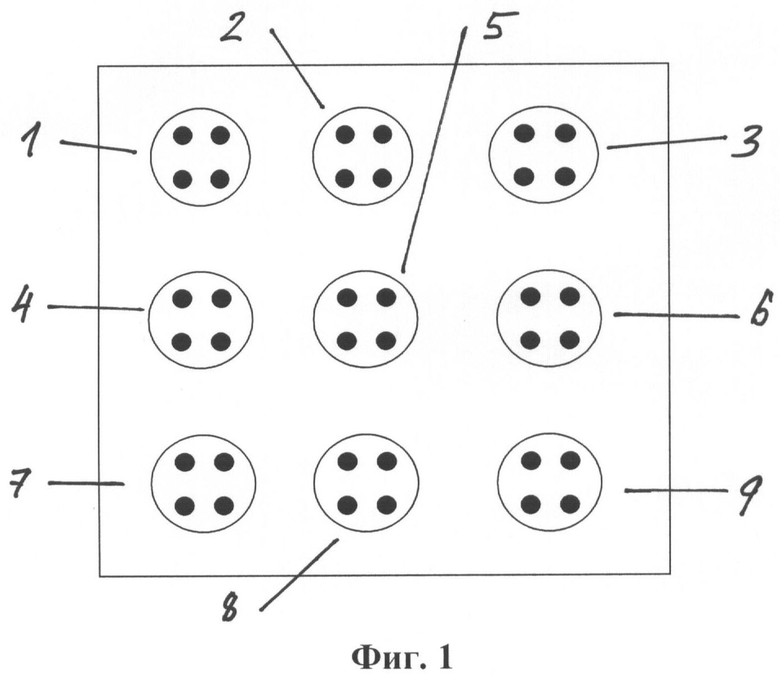
Фиг.1. Изображение расположения кластеров на рабочей зоне мирочипа, где: поз.1 и поз.8 относятся к положительному контролю, поз.2 относится к маркеру Shigella spp. и Enteroinvasive E.coli (EIEC), поз.3 и поз.7 относятся к отрицательному контролю, поз.4 относится к маркеру Salmonella spp., поз.5 относится к маркеру Campylobacter jejuni, поз.6 относится к маркеру Proteus mirabilis, поз.9 относится к маркеру Klebsiella pneumoniae.
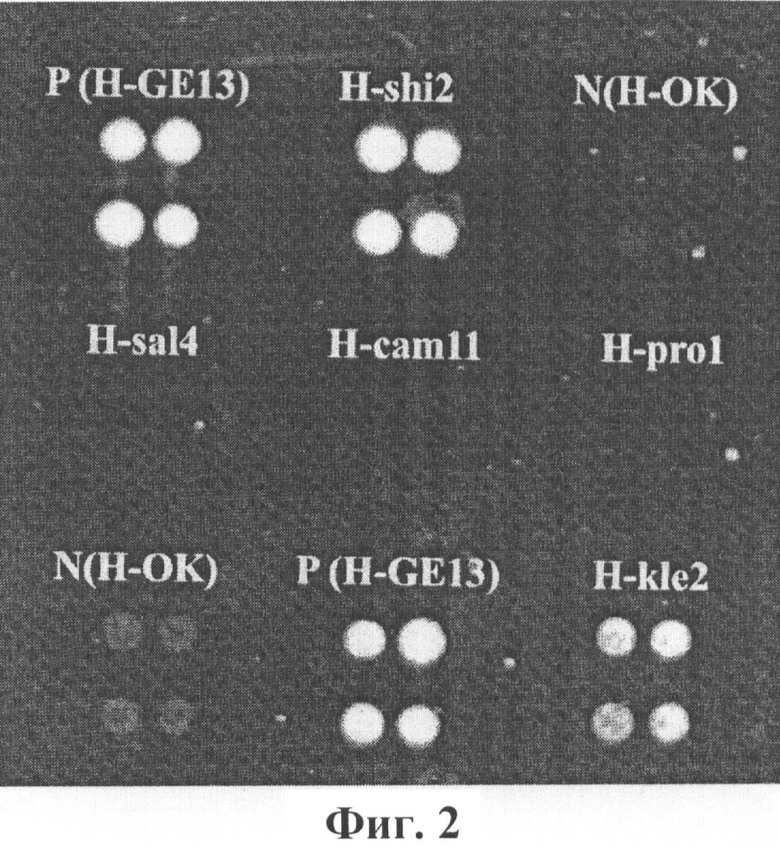
Фиг.2 Пример визуализации положительного результата теста на Shigella Spp., EIEC и Klebsiella pneumonaie. при идентификации образца, содержащего ОКИ.
Описание изобретения
Способ идентификации маркеров ОКИ, в общем случае, включает следующие стадии: (а) приготовление биологического микрочипа, заключающееся в модификации поверхности подложки микрочипа и в последующей иммобилизации дифференцирующих олигонуклеотидов, приведенных в таблице 3;
(б) осуществление выделения ДНК из клинического образца и использование рекомбинантной плазмиды охарактеризованной в таблице 1 для контроля процесса выделения ДНК;
(в) проведение мультиплексных симметричных полимеразных цепных реакций (ПЦР) образцов выделенных ДНК с использованием набора, содержащего биотин меченные праймеры, охарактеризованные в таблице №2,, а также, дополнительное проведение симметричных ПЦР с набором рекомбинантных плазмид, охарактеризованных в таблице 1 для осуществления положительного и отрицательного контроля процесса ПЦР;
(г) обработка продуктов амплифицированных ДНК экзонуклеазой фага лямбда;
(д) гибридизация амплифицированного меченого продукта на биочипе с образованием совершенных и несовершенных дуплексов и обеспечение положительного и отрицательного контроля процесса гибридизации;
(е) проведение реакции формирования комплекса с флуоресцентно-меченным стрептавидином;
(ж) регистрация и интерпретация результатов гибридизации путем сравнения полученной картины с шаблонами;
Выбор маркеров.
В процессе изучения способов диагностики ОКИ и известных диагностических биочипов [1-6] было просмотрено большинство известных решений, связаны с выбором известных маркеров для диагностики ОКИ.
К неочевидному решению в рамках настоящего изобретения можно отнести выбор перечня маркеров, с помощью которых осуществляют идентификацию более широкого круга патогенных микроорганизмов, чем в известных патентах аналогах. Перечень маркеров, составлен таким образом, что он устраняет недостатки известных изобретений. В перечне, в совокупности с известными маркерами генов, входящих в группу Shigella spp., EIEC, Salmonella spp., Campylobacter jejuni, Klebsiella pneumoniae [6], включен дополнительный маркер гена из Proteus mirabilis. Выбор Proteus mirabilis неочевиден, поскольку он используется в основном для идентифицирования урогенитальных инфекций в диагностических системах основанных на идентификации Mycobacterium, Mycoplasma, Legionella, Salmonella, Chlamydia, Campylobacter, Proteus mirabilis, Enterococcus, Enterobacter, Е. coli, Pseudomonas group I, Neisseria gonorrhoeae [8, 9]. Включение в совокупность маркеров маркер гена из Proteus mirabilis позволяет реализовать техническую задачу изобретения, связанную с повышением эффективности диагностики, одновременно, быстро и с минимальными затратами диагностировать в одном анализе от 1 до 5 маркеров ОКИ, основанном на идентификации участков ДНК, специфичных для источников ОКИ.
Выбор специфических олигонуклеотидов для мультипраймерной ПЦР.
При выборе последовательности для специфических праймеров ПЦР выполняли два условия.
Первое условие относилось к выбору участка фрагмента ДНК с новой последовательностью. При поиске новых фрагментов отслеживалось, чтобы комплементарность выбранного нового фрагмента по отношению к известному фрагменту из уровня техники была ниже 80-70%. Причем поиск таких последовательностей велся в более широкой области, чем диагностика ОКИ.
В процессе исследования новизны разработанных праймеров не было найдено патентов и заявок на изобретения, в которых была комплементарность между новыми заявляемыми и известными праймерами составляла 100%.
Праймеры выбраны с учетом требований высокой специфичности к консервативным участкам генов исследуемых объектов Shigella spp., EIEC, Salmonella spp., Campylobacter jejuni, Klebsiella pneumoniae Proteus mirabilis, что позволяет избирательно амплифицировать фрагменты ДНК патогенных микроорганизмов непосредственно из выделенного биологического образца. При выборе праймеров учитывают требования, стандартно применяемые к праймерам, в которые входят: отсутствие высокостабильных вторичных структур, малое расхождение температур плавления, не превышающее 2-3°C, а также выбор длины праймеров в диапазоне от 15 до 35 н.о [7].
Для составления праймеров использовались последовательности, характерные для идентифицируемых микроорганизмов и представленные в базе данных нуклеотидных последовательностей GenBank (http://ncbi.nlm.nih.gov), в которых выбирались наиболее консервативные участки, не используемые ранее и неочевидные для идентификации.
Каждая пара олигонуклеотидов для ПЦР, специфичных к одному из маркеров ОКИ, состоит из одного немеченного (не содержащего каких-либо меток, необходимых для последующего анализа и интерпретации результатов) специфического олигонуклеотида из перечисленных в списке последовательностей с SEQ ID NO: 1, 4, 7, 8, 11, 14, 17 и меченого (содержащего одну или несколько меток, необходимых для последующего анализа и интерпретации результатов) специфического олигонуклеотида из перечисленных в списке последовательностей с SEQ ID NO: 2, 5, 8, 11, 14, 17. Ниже приведен перечень специфических нуклеотидов (таблица 2) используемых в рамках настоящего изобретения.
Выбор дифференцирующих олигонуклеотидов.
Дифференцирующие олигонуклеотиды выбирали с учетом требований высокой специфичности к консервативным участкам генов исследуемых ОКИ, что позволяет проводить избирательную гибридизацию фрагментов ДНК, амплифицируемых со специфическими олигонуклеотидами при помощи ПЦР непосредственно из биологических образцов, а также исходя из отсутствия высокостабильных вторичных структур, и имеющие длину от 15 до 35 н.о.
Допустимо использование олигонуклеотидов, гомологичных указанным в перечне последовательностей олигонуклеотидам не менее чем на 80%, предпочтительно, не менее чем на 90%, наиболее предпочтительно, не менее чем на 95%. Допустимо использование олигонуклеотидов, имеющих делеции или вставки одного или нескольких нуклеотидов.
Ниже приведен перечень дифференцирующих олигонуклеотидов (таблица №3) для диагностики ОКИ в состав которых входят и дифференцирующие праймеры положительного и отрицательного контроля.
Синтез зондов и праймеров
Олигонуклеотиды синтезируют на автоматическом синтезаторе (например, модель "Gene Assembler" фирмы Pharmacia) стандартным амидофосфитным методом. Дифференцирующие Олигонуклеотиды содержат 5'- или 3'-концевую группу, обеспечивающую их иммобилизацию на поверхности чипа, например, 5'-концевую фосфатную группу, которую вводят при помощи химического фосфорилирования в процессе синтеза. Один из пары специфических олигонуклеотидов для проведения ПЦР модифицируют по 5'-концу меткой, которую выбирают из группы, состоящей из: каталитической, лигандной, флуоресцентной и радиоактивной, которую вводят химическими методами или энзиматически.
Выделение ДНК и подготовка материала для амплификации.
Выделение ДНК проводят из биологического образца, содержащего ОКИ с использованием общепринятых методик, основанных, например, на щелочном лизисе, использовании детергентов, протеиназы К, экстракции фенолом, хлороформом и/или смесью фенол/хлороформ, либо очистку на диатомовой земле или с помощью диоксида кремния.
Положительные и отрицательные контроли. В рамках данного изобретения существует несколько уровней контроля процесса диагностики. На этапе выделения ДНК в выделенную смесь добавляют внутренний контроль выделения ДНК. Для формирования функции контроля процесса амплификации используют положительный и отрицательный контроль. В качестве положительного контроля используют смесь рекомбинантных плазмид название которых приведено в таблице 1. Конструкция каждой плазмиды содержит клонированный фрагмент одного из маркерных генов входящего в группу Shigella spp. и Enteroinvasive E.coli (EIEC), Salmonella spp., Campylobacter jejuni, Proteus mirabilis, Klebsiella pneumoniae. Длина ПНР-фрагментов лежит в пределах от 82 до 138 п.н. Отрицательный контроль, для последующей проверки оптимальных условий проведения процедуры мультипраймерной ПЦР, выполнен в виде плазмиды с клонированным фрагментом гена GE 13 длиной 160 п.н.
Кроме этого на микрочипе иммобилизованы дифференцирующие олигонуклеотиды выполняющие роль положительного и отрицательного контролей прохождения реакции гибридизации (см. пример №).
Таким образом, в рамках данного изобретения обеспечивается полный контроль за всеми этапами процесса диагностики.
Выбор метки
Известно большое число патентов, в которых рассматриваются разные типы меток для построения биологических чипов. Каталитическую молекулу выбирают из группы, включающей гемин, цианкобаламин или флавин. Лигандную молекулу выбирают из группы, включающей биотин, диоксигенин или динитробензол. Флуоресцентную молекулу выбирают из группы, включающей FAM, TAMRA, Су3, Су5, Су7, R6G, R110, ROX или JOE.
Амплификация ДНК при помощи ПЦР
Фрагменты ДНК типичных последовательностей ОКИ, получают в реакции симметричной ПЦР с использованием высокоспецифичных праймеров (см. пример 4).
Условия ПЦР.
Общее количество пар специфических олигонуклеотидов может варьировать от 1 до 5 в сочетаниях для идентификации маркеров ОКИ. В состав ПЦР-смеси входит от 1 ед. до 20 ед., активности Taq-полимеразы на 1 мкл. В состав ПЦР смеси могут входить реагенты, повышающие специфичность и эффективность ПЦР, например, такие как бетаин, эктаин и его производные, а также трегалоза, диметилсульфоксид, глицерин.
Концентрация олигонуклеотидов в ПЦР может составлять от 0,05 мкМ до 0,3 мкМ, оптимально от 0,1 мкМ до 0,25 мкМ.
Объем реакционной смеси может составлять от 5 мкл до 200 мкл, оптимально от 20 мкл до 50 мкл. Реакцию можно проводить под минеральным маслом или без него, в зависимости от конструкции ДНК-амплификатора. ПЦР проводят с использованием ДНК амплификатора (например, «Терцик», ДНК-технология, Россия или «Mastercycler», Eppendorf, Германия) в условиях, приведенных в Таблице 4.
Обработка ПЦР продуктов экзонуклеазой фага лямбда.
Один из пары праймеров для ПЦР содержит фосфатную группу на 5'-конце. Это необходимо для того, чтобы цепь фрагмента ДНК, которая синтезируется в ходе ПЦР с этим праймером, эффективно гидролизовалась экзонуклеазой фага лямбда. Поскольку второй из пары праймеров на 5'-конце содержит биотин или флуоресцентную метку, эта цепь ДНК защищена от деградации экзонуклеазой. Это позволяет после обработки продуктов ПЦР экзонуклеазой фага лямбда получать одноцепочечную ДНК, меченную на 5'-конце биотином или флуоресцентной меткой. Гибридизация с одноцепочечной ДНК проходит более эффективно, что позволяет повысить чувствительность и воспроизводимость диагностики. Для получения одноцепочечной ДНК часто используют асимметричную ПЦР. Метод получения одноцепочечной ДНК с помощью асимметричной ПЦР требует большого количества циклов ПЦР и до 50-кратного избытка одного из праймеров, что приводит к увеличению времени проведения ПЦР и увеличивает расходы на проведение реакции. Применение экзонуклеазы фага лямбда для получения одноцепочечной ДНК позволяет использовать стандартную симметричную ПЦР, которая более эффективна и экономична как по количеству реагентов, так и по затрачиваемому времени. Для проведения гидролиза продуктов ПЦР используют от 2 до 20 единиц активности на 1 мкл. экзонуклеазы фага лямбда. Время обработки составляет от 30 до 90 мин. Более предпочтительно использовать 10 единиц фермента и проводить реакцию 60 минут при температуре 37°C.
Проведение гибридизации.
Меченые продукты ПЦР гибридизуются с ДНК-зондами (дифференцирующими олигонуклеотидами), иммобилизованными на биочипе, в специально подобранном буфере (см. пример 3).
Проведение пероксидазной реакции
В случае, если продукты ПЦР мечены биотином, для дальнейшей идентификации необходимо провести реакцию с коньюгатом стрептавидин-пероксидаза или стрептавидин-фосфатаза (см. пример №).
Структура биочипа
Биологический микрочип в одном из вариантов, который включает, но не ограничивает других вариантов, представляет собой стеклянную пластину следующих размеров: длина - 76,0±0,1 мм; ширина - 26,0±0,1 мм; толщина - 1±0,1 мм. На поверхности биологического микрочипа в определенном порядке расположены 9 кластеров иммобилизованных олигонуклеотидов (см. Фиг.1). В пяти кластерах иммобилизованы олигонуклеотиды, соответствующие последовательностям ДНК диагностируемых возбудителей - Shigella spp. и Enteroinvasive E.coli (EIEC), Salmonella spp., Campylobacter jejuni, Proteus mirabilis, Klebsiella pneumoniae (H-shi, H-sal, H-cam, H-pro, H-kle). В двух кластерах с индексом «P» содержится ковалентно связанный олигонуклеотид H-GE13, предназначенный для проверки всех реакций набора и однозначной ориентации биологического микрочипа. Два кластера с индексом «N» содержат олигонуклеотид H-17, который не соответствует диагностируемым последовательностям и выполняет роль отрицательного контроля гибридизации. Расположение и предназначение рабочих зон на поверхности биочипов соответствует схеме расположения ДНК-зондов. Дифференцирующие олигонуклеотиды - зонды нанесены в кластере в четырех или девяти повторах.
Предпочтительной формой мультичипа является плоский прямоугольный элемент, выполненный из твердого или гибкого материала разной толщины. Толщина несущего элемента может составлять от 0,05 мм до 5 мм. Несущий элемент чипа выполнен из материалов входящих в группу, состоящую из: полимеров, стекла, металлов, керамики или их комбинаций. Полимеры выбирают из группы состоящей из: полиметилметакрилата, полибутилметакрилата, поливинилхлорида, поликарбоната, сополимеров метилметакрилата и/или сополимеров бутилметакрилата с другими мономерами, такими, как стирол, акрилонитрил.
Поверхность несущего элемента может быть модифицирована нужным образом или покрыта каким-либо материалом, способным иммобилизовать олигонуклеотиды.
На фиг.2 приведена гибридизационная картина, полученная при сканировании биочипа, в которой приведен пример визуализации положительного результата теста на Shigella Spp., EIEC и Klebsiella pneumonaie. при идентификации образца, содержащего ОКИ.
Идентификация мультичипа
Идентификатор биочипа может включать в себя код номера партии и/или даты изготовления, информацию о перечне размещенных дифференцирующих олигонуклеотидов. Идентификатор может быть выполнен в виде штрих-кода или элемента с магнитной записью информации.
Обращение к уникальному коду биочипа обеспечит быстрый поиск информации в базах данных о всех образцах при параллельном скрининге ОКИ.
Интерпретация данных анализа
Интенсивность сигнала образующихся совершенных или несовершенных дуплексов внутри каждой группы кластеров существенно выше интенсивности сигнала отрицательных контролей, что позволяет проводить надежную идентификацию ОКИ. Детектирование и/или идентификацию ОКИ осуществляют путем сравнительного анализа уровня сигналов исследуемого образца, положительного и отрицательного контролей Интерпретацию результатов проводят исходя из гибридизационной картины, которую получают, используя экспериментальную установку, включающую, например, сканер, компьютер и специальное программное обеспечение. Для считывания данных с микрочипа, более предпочтительно использовать для интерпретации результатов анализа аппаратно-программный комплекс - чип-детектор «Флуоген-2» изготовляемый фирмой ММТех, Россия.
Интерпретацию результатов проводят следующим образом. Интенсивности флуоресцентных или колориметрических сигналов в каждой группе ДНК-зондов, в которой содержатся олигонуклеотиды, видоспецифичные для одного из 9 маркеров ОКИ, с помощью специального программного обеспечения сравнивают с группами зондов положительного (максимальная интенсивность сигнала) и отрицательного (минимальная интенсивность сигнала) контроля. В результате образуется гибридизационная картина, различная для групп положительного контроля, отрицательного контроля и для кластеров, характеризующих маркеры ОКИ, что позволяет однозначно интерпретировать полученные данные.
Набор для диагностики
Другим объектом изобретения является набор для идентификации маркеров ОКИ, включающий:
а) по меньшей мере, один биочип для проведения разовой диагностики, содержащий не менее пяти дифференцирующих олигонуклеотидов, два из которых служат в качестве положительного и два в качестве отрицательного контролей и представленных в SEQ SEQ ID NO: 3, 6, 9, 12, 15, 18, 19, соответственно;
б) специфические олигонуклеотиды, используемые в качестве праймеров ПЦР для идентификации маркеров ОКИ, последовательности которых представлены в SEQ ID NO: 1, 2, 4, 5, 7, 8, 10, 11, 13, 14, и специфический олигонуклеотиды, представленный в SEQ ID NO: 16, 17 которые комплементарны дифференцирующему олигонуклеотиду положительного контроля SEQ ID NO: 18, размещенному на биочипе в кластерах положительного контроля.
в) реагенты, которые выбирают из группы, состоящей из: реагентов для подготовки образца, реагентов для проведения мультипраймерной симметричной ПЦР, реагентов для проведения гибридизации или их комбинаций и экзонуклеазы фага лямбда.
Набор дополнительно содержит наклейки с названием торговой марки и штрихкодом, в котором указан номер серии и дата выпуска диагностической системы.
Примеры
Ниже приведены примеры, которые включают, но не ограничивают объем изобретения.
Пример 1. Приготовление поверхности биочипа (модификация поверхности)
Матрицу, содержащую аминогруппы с плотностью 1-2 групп/нм2, изготавливают с применением раствора 3-аминопропилтриэтоксисилана (АПТЭС) в различных растворителях в зависимости от типа подложек. Например, стеклянные подложки инкубировали в течение часа в растворе 3-аминопропилтриэтоксисилана в ацетоне или в этаноле. Концентрацию АПТЭС варьировали от 0,1 до 10% по объему. Если реакцию проводят в ацетоне, стекла трижды промывали ацетоном по 5 минут, затем этанолом 2 раза по 3 минуты и сушили при температуре 120-130°C в течение 20 минут.
Пример 2. Пришивка олигонуклеотидных зондов к поверхности амино-модифицированного стекла.
Реакционную смесь, содержащую 1 мкМ олигонуклеотида и 60 мМ гидрохлорида 1-этил-3-(3-диметиламинопропил)-карбодиимида в 0,1 М 1-метилимидазоле, pH 7, наносили на поверхность матрицы роботом (Xpress Lane, США). Объем капель каждого зонда составляет не менее 0,005 мкл. Матрицы с нанесенными олигонуклеотидами помещали в герметичную емкость, выдерживали при комнатной температуре в течение 1 часа во влажной атмосфере и промывали на шейкере 0,5 мМ раствором NaCl в течение содержащим 0,1% SDS в течение 10 минут, затем дистиллированной водой. Биочип высушивали и хранили при температуре от 4 до 8°C. Концентрация олигонуклеотида в объеме капель каждого зонда составляет от 0,5 до 100 мкмоль/л.
В качестве зондов использовали олигонуклеотиды, модифицированные фосфатной группой (p) по 5'-положению ID SEQ No: 59, 68, 90 и 91. Олигонуклеотиды наносили кластерами по 4 точки.
Пример 3. Выделение ДНК из исследуемого материала.
В промаркированные пробирки добавляют по 1 мл физиологического раствора и по 0,2-0,3 г фекалий (что приблизительно соответствует объему 200-250 мкл). Для этого используют одноразовые лопатки или наконечники с антиаэрозольными фильтрами. Тщательно перемешивают содержимое на вортексе до образования гомогенной суспензии. Осаждают грубодисперсную фракцию центрифугированием на микроцентрифуге в течение 2 минут при 2000 об/мин. Надосадочную жидкость переносят в новую пробирку и центрифугируют при максимальных оборотах (12000 об/мин.) в течение 3 минут. После удаления надосадочной жидкости к бактериальному осадку добавляют 1 мл физиологического раствора, суспендируют на вортексе 30-60 сек. и затем осадают центрифугированием при 12-13 тыс. об/мин. После окончательной отмывки осадок с исследуемым материалом ресуспендируют в 300 мкл физиологического раствора.
В промаркированные пробирки добавляют по 400 мкл лизирующего буфера и 5 мкл положительного контрольного образца KB.. Лизирующий буфер готовят на деионизованной воде на основе Трис-HCl, pH 6,8 - 0,05 М, гуанидинтиоцианата - 3,13 М (фирма «Amresco», США) GTC, ЭДТА - 0,025 М (фирма «Sigma», США) и саркозила - 1%. (фирма «AppliChem», США). Затем в каждую пробирку добавляют по 100 мкл исследуемого материала, в пробирку отрицательного контроля выделения ОКВ добавляют 100 мкл деионизованой воды. Перемешивают и инкубируют при 65°C в течение 5 минут. В каждую пробирку добавляют 20 мкл сорбента C, перемешивают и инкубируют при комнатной температуре в течение 10 мин.
После этого пробирки центрифугируют в течение 20 сек при 10 тыс. об/мин, и удаляют надосадочную жидкость. В каждую пробирку к осадку добавляют по 750 мкл первого буфера выполненного на основе Трис-HCl, pH 6,8 - 0,0585 М, гуанидингидрохлорида - 2,34 М (фирма «Fluka», США), и ЭДТА - 0,0243 М (фирма «Sigma»), ресуспендируют осадок перемешиванием на вортексе в течение 30-60 сек. Центрифугируют в течение 20 сек при 10 тыс. об/мин, и удаляют надосадочную жидкость.
В каждую пробирку к осадку добавляют по 750 мкл второго буфера выполненного на основе Трис-HCl (1 М Трис-HCl, pH 8,0 с конечной концентрацией 0,05 М), ресуспендируют осадок перемешиванием на вортексе в течение 30 сек. Затем центрифугируют в течение 20 сек при 10 тыс. об/мин, и удаляют надосадочную жидкость. Повторяют данную операцию дважды.
Подсушивают осадки в термостате при 55°C в течение 5 минут (крышку пробирки оставить открытой). К осадкам добавляют по 50-100 мкл буфера для элюции, выполненного на основе Chelex 100 - 5% (фирма «BIO-RAD» США) и Трис-HCl, pH 8,0 - 0,05 М. Перемешивают на вортексе и инкубируют при 55°C в течение минут. Для более эффективной элюции ДНК, встряхивают пробирку каждые 2 минуты. Затем пробирки центрифугируют в течение 1,5-2 мин при 10-12 тыс. об/мин. Переносят надосадочные жидкости в чистые промаркированные микропробирки. Хранят выделенные ДНК при температуре минус 20°C в течение года. Допустимо хранение при комнатной температуре (от 18 до 25°C) не более 24 ч, при температуре от 4 до 8°C - не более 7 дней. Полученную ДНК используют для проведения этапа ПЦР.
Пример 4. Мультипраймерная полимеразная цепная реакция.
Приготовляют «ПЦР-смесь», исходя из количества анализируемых образцов плюс две пробирки для «-» отрицательного и «+» положительного контролей на ПЦР. На каждую пробу вносят по 11 мкл 2-кратного реакционного буфера, 5 мкл биологически активных элементов в виде композиции олигонуклеотидных праймеров SEQ ID NO: 1, 2, 4, 5, 7, 8, 10, 11, 13, 14, 16, 17 приведенных в таблице №2 и 1 мкл Taq ДНК-полимеразы (фирма «Fermentas», Латвия). Аккуратно перемешивают пипетированием. 2х реакционный буфер готовят на деионизованной воде на основе Трис-HCl, (NH4)2SO4, MgCl2, добавляют раствор дНТФ. Состав: 1 М Трис-HCl, pH 8,8-0,16 М; (NH4)2SO4 - 0,032 М (фирма «Sigma», США); MgCI2 (фирма «Sigma», США) - 0,004 М; смесь дНТФ (фирма «Силекс», Россия) - 0,004 М; твин 20 (фирма «Amresco», США) - до конечной концентрации 0,04%.
В ранее промаркированные микропробирки для ПЦР вносят по 2 капли масла для ПЦР (~20-25 мкл), а затем по 15 мкл «ПЦР-смеси». В пробирки для исследуемых проб вносят пипеткой с отдельными наконечниками по 5 мкл ДНК исследуемого образца. В пробирку маркированную знаком «+» (положительный контроль ПЦР) вносят 5 мкл положительного контрольного образца ПК. ПК-положительный контрольный образец-смесь рекомбинантных плазмид, полученных на основе pUC18 и содержащих последовательность маркерных генов каждого возбудителя в концентрации 5 пг/мкл (или 1,5×106 молекул ДНК в 1 мкл). В пробирку, маркированную знаком «-» (отрицательный контроль ПЦР), вносят 5 мкл отрицательного контрольного образца ОК (добавляется последним). OK - отрицательный контрольный образец - рекомбинантная плазмида, полученная на основе pUC18 и содержащая последовательность маркера GE13 в концентрации 50 пг/мкл (или 15×106 молекул ДНК в 1 мкл). Центрифугируют микропробирки 10-15 сек при 10-12 тыс. об/мин для осаждения капель жидкости, при этом ПЦР-масло должно полностью покрыть ПЦР-смесь.
Подготовленные для проведения реакции пробирки перенести в ДНК-амплификатор, и запускают программу амплификации в соответствии с режимами, приведенными в таблице 4.
После окончания программы амплификации, добавляют к 20 мкл каждой пробы 1 мкл экзонуклеазы (фирма «Fermentas», Латвия) в буферном растворе в концентрации 2 ед. активности/мкл., аккуратно перемешивают пипетированием и инкубируют 1 час при 37°C. Добавляют к каждой пробе 2 мкл стоп-раствора, который готовят на деионизованной воде на основе ЭДТА (фирма «Sigma», США,) в концентрации 0,020 М. Перемешивают и осаждают продукты ПЦР и инкубируют пробу 10 мин при 80°C. При необходимости после этой стадии пробу можно хранить при минус 20°C в течение 3-х месяцев.
Пример 3. Гибридизация продуктов ПЦР на биочипах.
Добавляют в каждую микропробирку с ПЦР-фрагментами, полученными на этапе ПЦР, 2,5 мкл 20×SSC, 2 мкл 1% SDS и перемешивают пипетированием. Полученную смесь наносят на середину рабочей зоны микрочипа и накрывают пленкой размером 16×21 мм так, чтобы под ней не осталось пузырьков воздуха. Использование пленки позволяет значительно снизить расход реактивов. Помещают микрочип во влажную камеру (например, в чашку Петри со смоченным дистиллированной водой бумажным фильтром) и инкубируют 1 час в воздушном термостате при температуре 44°C. По окончании инкубации смывают пленку с микрочипа и промывают чип на орбитальном шейкере следующими растворами:
2×SSC, 0,1% SDS - 1 раз 5 минут;
1×SSC, 0,1% SDS - 1 раз 5 минут;
0,5×SSC - 2 раза по 5 минут.
По окончании промывки микрочип подсушивают при комнатной температуре.
Пример 4. Реакция образования комплекса с флуоресцентно-меченным стрептавидином.
Формируют реакционную смесь, содержащую 1×SSC, 10 мг/мл БСА (бычий сывороточный альбумин, фирма «Amresco», США) и 0,1×ФМС (фирма «Thermo Scintific», США) из расчета 10 мкл на микрочип. Рекомендуется приготовить этот реактив с небольшим запасом. Например, для приготовления 210 мкл реакционной смеси (достаточно для обработки 20 микрочипов) необходимо к 10,5 мкл 20×SSC добавить 157,5 мкл бидистиллированной воды, 21 мкл БСА. Перемешивают реакционную смесь и добавляют 20 мкл ФМС. Наносят 10 мкл приготовленной реакционной смеси на рабочую зону микрочипа, накрывают пленкой размером 16×21 мм и помещают на 1 час во влажную камеру при температуре 28°C. После инкубации проводят отмывку чипа на орбитальном шейкере: в растворе 1×SSC - 2 раза по 5 минут и в растворе 0,25×SSC 1 раз 5 минут.
Пример 5. Учет результатов диагностики.
Для считывания данных с микрочипа, используют аппаратно-программный комплекс, предназначенного для выявления наличия или отсутствия на микрочипе связавшихся с ним специфичных, флуоресцентно-меченых последовательностей ДНК. В качестве одного из вариантов используют чип-детектор «Флуоген-2» (ММТех, Россия).
Интерпретация результатов анализа осуществляется компьютерной программой «Gene Inspector» на основе заданного алгоритма. Алгоритм учитывает величины сигналов в зонах положительных и отрицательных контролей, а также зоны анализируемых инфекций. На чипе может наблюдаться сигнал в зоне отрицательного контроля, величина которого незначительно варьирует в зависимости от точности соблюдения условий гибридизации и отмывок, указанных в инструкции. Результат считается положительным, в случае, если значение разницы сигналов в зоне анализируемой инфекции и отрицательного контроля больше нуля.
Набор обеспечивает выявление 102-103 копий ДНК каждого из возбудителей Shigella spp. и Enteroinvasive E.coli (EIEC), Salmonella spp., Campylobacter jejuni, Proteus mirabilis и Klebsiella pneumoniae в 1 мл пробы.
Литература
1. Schrenzel J. et al. Analytical chip with an array of immobilized specific recognition elements for the determination of clinically relevant bacteria and analytical method based thereon US Patent Application 20070015151 (January 18, 2007).
2. Chang; Tsung Chain; et al. Method for rapidly identifying bacteria and kit thereof. 20060228718 October 12, 2006
3. Peters, et al. Customized micro-array construction and its use for target molecule detection. US patent №7,364,898 April 29, 2008
4. Jacobs; Alice A.; et al. Clinically intelligent diagnostic devices and methods. US Patent Applic. №20060240453 October 26, 2006
5. Persson; Soren; et al. Diagnostics of diarrheagenic escherichia coli (dec) and shigella spp. US Patent Applic №20060194206 August 31, 2006
6. Кверк С. ОБНАРУЖЕНИЕ И ИДЕНТИФИКАЦИЯ КИШЕЧНЫХ БАКТЕРИЙ Заявка на изобретение РФ №2004114259 (2002.10.01).
7. Грановский И.Э. ДИФФЕРЕНЦИРУЮЩИЙ И СПЕЦИФИЧЕСКИЙ ОЛИГОНУКЛЕОТИД ДЛЯ ИДЕНТИФИКАЦИИ ПОСЛЕДОВАТЕЛЬНОСТЕЙ ДНК ТРАНСГЕННЫХ РАСТЕНИЙ В ПИЩЕВЫХ ПРОДУКТАХ, СПОСОБ ИДЕНТИФИКАЦИИ ТРАНСГЕННЫХ ПРОДУКТОВ, БИОЧИП, КОМБИНАЦИЯ ОЛИГОНУКЛЕОТИДОВ (ВАРИАНТЫ) И НАБОР ДЛЯ ОСУЩЕСТВЛЕНИЯ ЭТОГО СПОСОБА. Заявка RU №2007134194/13 (20.03.2009).
8. Hogan Nucleic acid probes and methods for detecting Proteus mirabilis. US patent №5,683,876 November 4, 1997.
9. Hogan, et al. Oligonucleotide probes for the detection and/or quantitation of non-viral organisms US patent №7,138,516 November 21, 2006.






Изобретение относится к области молекулярной биологии и может быть использовано в диагностических исследованиях, направленных на выявление возбудителей острых кишечных инфекций (ОКИ). Предложен набор дифференцирующих олигонуклеотидов (зондов), обеспечивающий возможность определения в биологическом образце ДНК патогенных микроорганизмов, вызывающих ОКИ, и их видовой идентификации, где указанные микроорганизмы относятся к группе, включающей Shigella spp. и Enteroinvasive E.coli (EIEC), Salmonella spp., Campylobacter jejuni, Proteus mirabilis, Klebsiella pneumoniae. Описано конструирование биочипа с иммобилизованным на нем набором зондов по изобретению, предназначенного для проведения экспресс-анализа возбудителей ОКИ в клинических образцах и материале, полученном из объектов окружающей среды. 2 н.п. ф-лы, 4 табл., 2 ил., 5 пр.
1. Набор дифференцирующих олигонуклеотидов для экспресс-анализа видовой идентификации возбудителей острых кишечных инфекций, последовательность которых определена в Перечне последовательностей (SEQ ID NO: 3, 6, 9, 12, 15, 18, 19).
2. Биочип для экспресс-анализа видовой идентификации возбудителей острых кишечных инфекций, представляющий собой подложку с набором иммобилизованных на ней олигонуклеотидов, охарактеризованных по п.1.
| RU 2004114259 A, 01.10.2002 | |||
| CHIZHIKOV V | |||
| ET AL., Appl | |||
| Env | |||
| Microdiol., 67 (7), 3258-32-63, 2001 | |||
| US 20060240453 A, 26.10.2006. |
Авторы
Даты
2014-03-20—Публикация
2010-03-31—Подача