Область техники
Изобретение относится к области биотехнологии, молекулярной биологии и генной инженерии, а именно к способу идентификации последовательностей ДНК генетически модифицированных источников и пищевых продуктов на их основе, включающей этапы амплификации ДНК и последующей гибридизации амплифицированной ДНК на биологическом микрочипе. Изобретение также раскрывает состав высокоспецифичных ДНК-зондов и олигонуклеотидов для ПЦР, набор реактивов для идентификации.
Уровень техники
Разработка диагностики генетически модифицированных источников (ГМИ) в продуктах питания в настоящее время является актуальной задачей. Директивой Европейского Парламента и Совета (ЕС) от 22.09.2003 №1829/2003 о генетически модифицированной пище и кормах [1] с апреля 2004 года введены новые правила маркировки в странах ЕЭС, по которым установлен пороговый уровень ГМИ, равный 0,9%. Введение 0,9% порогового уровня связано с чувствительностью метода определения ГМИ и случайным попаданием ГМИ в пищевые продукты. Обязательная маркировка пищевых продуктов из ГМИ введена и в России.
Известны способы для определения ГМИ в пищевой продукции. Один из способов основан на применении иммуноферментного анализа с использованием соответствующих антител к белкам, входящим в состав генетически модифицированных растений [2].
Более стабильным и распространенным является способ идентификации, основанный на анализе ДНК, входящих в состав трансгенных растений с использованием полимеразной цепной реакции (ПЦР) [3, 4, 5, 6, 7].
В настоящее время в Российской Федерации действует ГОСТ 52173-2003 по определению ГМИ в пищевых продуктах и растительном сырье методом ПЦР [8]. В процессе проведения ПЦР используют выделенные из растительного сырья ДНК и пары синтетических праймеров. После проведения реакции амплификации продукты реакции разделяют методом гель-электрофореза и детектируют путем окрашивания их в геле флуоресцентным красителем. Амплифицированные фрагменты ДНК, содержащие флуоресцентную метку, идентифицируют либо после проведения реакции амплификации, либо в режиме реального времени путем измерения интенсивности флуоресцентного сигнала.
В последнее время все более широко применяют биочипы, которые позволяют упростить процедуру идентификации ГМИ [9, 10, 11]. В настоящее время действует ГОСТ 52174-2003 по определению ГМИ в пищевых продуктах и растительном сырье с помощью микрочипов [12]. Вопросы повышения точности и надежности определения ГМИ с помощью биочипов обычно решаются с помощью усовершенствования какой-либо одной процедуры в многоступенчатом процессе создания биочипа или в процессе гибридизации и сканирования полученных результатов. Тем не менее, известные технические решения обладают рядом недостатков, которые связаны либо с неудовлетворительным выбором участков генов, либо с выбором параметров мультипраймерной ПЦР, либо с конструкцией биочипа, например, с расположением кластеров на рабочем поле биочипа.
К одному из недостатков известных методов идентификации ГМИ относится достаточно узкий набор генетических маркеров и соответствующих праймеров для определения наличия маркеров трансгенных растений в процессе идентификации ГМИ. В одном из вариантов количественной идентификации ГМИ используют маркеры RRS, Bt-11, 35S CMV, а в качестве референсного маркера используют ген пектина [3]. В другом варианте [14] используют маркеры HMG, RRS02, в заявке [13] маркеры EPSPS, cry1A(b), cry9C и PAT.
Известно изобретение [15], в котором для определения наличия ГМИ используют от 3 до 10 пар праймеров для проведения мультипраймерной процедуры ПЦР. Генетические маркеры выбирают из группы P35S, TNOS, PNOS, NPTII, GUS, ген EPSPS дикого типа, ген EPSPS модифицированного типа, СР4, vignatrypsase CpTI и ген antibiotic peptide shiva. Продукты амплификации гибридизуют на мембране, снабженной специфическими зондами, и проводят детекцию результатов гибридизации. К недостатку изобретения можно отнести тот факт, что в перечне маркеров нет маркеров положительного и отрицательного контроля.
Выбор параметров мультипраймерной ПЦР в значительной степени определяет эффективность и стабильность диагностики. Известны работы, в которых для повышения точности в процедуру включают дополнительную ферментативную обработку. Известно изобретение [17], в котором описан способ амплификации ДНК с использованием композиции реагентов, которая содержит в качестве основного компонента термостабильный фермент, проявляющий активность ДНК-полимеразы, а в качестве минорного компонента - термостабильный фермент из Archeoglobus fulgidus, проявляющий активность 3'-экзонуклеазы, но лишенный активности ДНК-полимеразы. Согласно изобретению, фермент способен взаимодействовать с Taq полимеразой в качестве корректирующего фермента для того, чтобы удалить ошибочно спаренные концы праймеров, которые приводят к преждевременным остановкам, а также чтобы освободить концы праймеров, которые более эффективно элонгируются полимеразой, исправить ошибки включения оснований и дать возможность полимеразе образовать протяженные продукты PCR. Использование указанной композиции повышает точность процесса амплификации по сравнению с использованием только основного фермента.
Известен способ [18], в котором изложен принцип обработки праймеров, полученных в результате ПЦР, с помощью экзонуклеазы лямбда или продуктом гена 6 фага Т7 в течение 15 мин при температуре 37°С. В качестве останавливающего агента используют раствор ЭДТА с концентрацией 10 mM. В данном изобретении не рассматриваются вопросы обработки экзонуклеазой продуктов, полученных в процессе мультипраймерной ПЦР, и факт обработки экзонуклеазой
Известен способ [19], в котором рассматривается одновременное проведение множественной амплификации нуклеиновых кислот и использование экзонуклеазы. Согласно изобретению, способ включает: (а) контакт нуклеиновой кислоты с одной или более парами праймеров в условиях, которые позволяют провести отжиг праймеров с нуклеиновой кислотой, причем каждый праймер имеет двухстороннюю структуру А-В, в которой часть А специфична для целевых последовательностей образца нуклеиновой кислоты, а часть В является постоянной последовательностью, общей для всех праймеров; (б) проведение первой реакции амплификации; (в) обработку двухсторонних праймеров или продуктов первой реакции амплификации с помощью экзонуклеазы; (г) осуществление контакта продуктов первой реакции с праймерами, содержащими часть В, в условиях гибридизации, и проведение второй реакции амплификации. В данном изобретении в качестве целевых последовательностей используют гены Bt176, Bt11, Mon810, Amp, Nos, 35S, IPC, Corn ref. В качестве фермента выбрана экзонуклеаза I. К недостатку способа можно отнести необходимость проведения двухстадийной ПЦР.
Известны наборы [20], которые выпускает ЗАО "БиоХимМак", в которые входят: реакционная смесь для детекции ГМИ (2×50), референсная смесь, калибраторы ДНК растительного гена и ГМИ, положительный контроль. В состав набора входит один из генов, входящих в группу Раундап Реди, Bt176 Максимайзер, Bt11, ЛибертиЛинк, ЙелдКард MON810, СтарЛинк, GA21, ITC 1507, LL (T25). С помощью наборов проводят количественный анализ ГМИ методом ПЦР в реальном времени (наборы GMO-Quant). Известно изобретение [15], которое описывает кит для детекции ГМИ, в состав которого входят праймеры и зонды для проведения мультиплексной ПЦР и гибридизации. Ближайший аналог заявленного изобретения в части набора, в который входят праймеры и реактивы для создания реакционных смесей, описан в методических указаниях МУК 4.2.2008-05 [16].
Наиболее близким аналогом в части способа, является изобретение [9], в котором для идентификации ГМИ используют генетические маркеры, выбираемые из группы: 35S - промоторного участка вируса мозаики цветной капусты, маркерного гена gus из бактерии Escherichia coli, терминаторного участка гена nos из Agrobacterium tumefaciens, маркерного гена nptII из транспозона Tn5, терминаторного участка гена ocs из Agrobacterium tumefaciens, которые включены в методические указания [16].
Способ идентификации трансгенных последовательностей ДНК в растительном материале и продуктах на основе такого материала [9] включает анализ ДНК при помощи полимеразной цепной реакции (ПЦР) и регистрацию результатов анализа, причем ПЦР проводят на препарате ДНК в виде мультиплексной реакции с получением флуоресцентно меченного продукта, при этом используют специфические пары праймеров, один из которых флуоресцентно мечен и присутствует в избытке по отношению ко второму немеченному праймеру, результаты ПЦР анализируют с помощью гибридизации на биочипе и регистрируют с помощью аппаратно-программного комплекса, позволяющего отображать флуоресценцию ячеек биочипа на экране компьютера. Однако изобретение содержит ограниченное число праймеров, а в состав набора не включены ферменты, позволяющие повысить эффективность анализа при использовании мультипраймерной симметричной ПЦР.
Техническая задача изобретения состоит в том, чтобы создать более совершенную диагностику ГМИ на основе нового способа с использованием нового биочипа, обладающего более совершенными характеристиками при невысокой стоимости скрининга ГМИ, и на основе набора для проведения диагностики, в состав которого входят новые праймеры, новый биочип и фермент для обработки продуктов ПЦР.
Другая задача состоит в увеличении достоверности и воспроизводимости получаемых результатов. Эта задача решается за счет применения в биологических чипах для диагностики ГМИ новых, более эффективных последовательностей праймеров и за счет разработки способа обработки продуктов мультипраймерной симметричной ПЦР ферментом, повышающим эффективность гибридизации, а также за счет размещения кластеров положительного контроля одновременно выполняющих роль идентификаторов границ рабочей зоны биочипа.
Сущность изобретения
Предлагаемый метод диагностики основан на проведении мультипраймерной симметричной ПЦР с использованием специфических праймеров, комплементарных последовательностям генов-маркеров ГМИ, и последующей гибридизации продуктов ПЦР на биочипе, содержащем оригинальный набор дифференцирующих олигонуклеотидов.
Одним из объектов изобретения является способ идентификации маркеров генетически модифицированных растений, включающий обеспечение образца с исследуемыми ДНК, прямых и обратных праймеров, специфичных к ДНК, принадлежащим к генам маркеров ГМИ, и биочипа, проведение мультипраймерной ПЦР в присутствии образца с исследуемыми ДНК и специфичными праймерами к указанным маркерам ГМИ, обработку продуктов ПЦР реакции ферментом, инкубацию ПЦР-продукта с дифференцирующими олигонуклеотидами, иммобилизированными на поверхности биочипа в условиях гибридизации, определение наличия ГМИ детекцией образованных гибридизационных комплексов на биочипе. В указанном способе биочип содержит более 6 типов дифференцирующих олигонуклеотидов, выбираемых из последовательностей, представленных в SEQ ID NO: 23-33, с учетом требований высокой специфичности к консервативным участкам генов ГМИ, входящих в перечень, приведенный в таблице 1. В качестве специфичных праймеров используют праймеры, последовательность которых выбирают из группы, состоящей из SEQ ID NO: 1-22, в качестве ПЦР используют мультипраймерную симметричную ПЦР, продукты которой предпочтительно в объеме 20 мкл обрабатывают предпочтительно 1 мкл раствора экзонуклеазы фага лямбда в концентрации от 5 до 20 единиц активности и инкубируют в течение от 30 мин до 90 мин, предпочтительно при температуре 37°С. В паре прямого и обратного праймеров, один праймер содержит метку, которую после гибридизации ДНК детектируют, регистрируют и используют для идентификации маркеров ГМИ, причем в качестве отрицательного контроля используют дифференцирующий олигонуклеотид SEQ ID NO: 36, частично комплементарный олигонуклеотиду SEQ ID NO: 8, а в качестве положительного контроля используют дифференцирующий олигонуклеотид SEQ ID NO: 34, которому комплементарен специфический олигонуклеотид SEQ ID NO: 35.
Другим объектом изобретения является специфический олигонуклеотид для использования в способе идентификации маркеров генетически модифицированных растений по п.п.1-4, который выбирают в качестве праймера симметричной мультипраймерной ПЦР и который характеризуется нуклеотидной последовательностью, выбранной из последовательностей, представленных в SEQ ID NO: 4, 7, 8, 11-14, 21, 22.
Следующим объектом изобретения является дифференцирующий олигонуклеотид в качестве зонда для идентификации маркеров генетически модифицированных растений для использования в способе идентификации ГМИ по п.п.1-4, который характеризуется нуклеотидной последовательностью, выбранной из последовательностей, представленных в SEQ ID NO: 24, 28, 29, 33.
Другим объектом изобретения является комбинация олигонуклеотидов для выявления маркера NOS для идентификации 3'-нетранслируемой области гена нопалинсинтазы, включающая олигонуклеотиды с последовательностями SEQ ID NO: 24, и SEQ ID NO: 3, и SEQ ID NO: 4.
Другим объектом изобретения является комбинация олигонуклеотидов для выявления маркера NPTII, кодирующего неомицин-фосфотрансферазу II, включающая олигонуклеотиды с последовательностями SEQ ID NO: 26, и SEQ ID NO: 7, и SEQ ID NO: 8.
Другим объектом изобретения является комбинация олигонуклеотидов для выявления маркера Lectin, представленного семейством генов Le1 из Glycine max, включающая олигонуклеотиды с последовательностями SEQ ID NO: 28 в качестве зонда и два специфических SEQ ID NO: 11 и SEQ ID NO: 12.
Другим объектом изобретения является комбинация олигонуклеотидов для выявления маркера Zein, представленного семейством генов zein из Zea Mays, включающая олигонуклеотиды с последовательностями SEQ ID NO: 29, и SEQ ID NO: 13, и SEQ ID NO: 14.
Другим объектом изобретения является комбинация олигонуклеотидов для выявления маркера Patatin, представленного семейством генов pat1 из Solanum tuberosum, кодирующего предшественник пататина, включающая олигонуклеотиды с последовательностями SEQ ID NO: 33, и SEQ ID NO: 21, и SEQ ID NO: 22.
Другим объектом изобретения является биочип для идентификации маркеров ГМИ, представляющий собой несущий элемент, на котором иммобилизованы дифференцирующие олигонуклеотиды в виде зондов, формирующих кластеры, характеризующийся тем, что на его поверхности иммобилизированы более 6 типов кластеров с дифференцирующими олигонуклеотидами, выбранными из SEQ ID NO: 23-33, при этом биочип содержит, по меньшей мере, один кластер, включающий олигонуклеотид положительного контроля, представленный в SEQ ID NO: 34, и, по меньшей мере, один кластер, включающий олигонуклеотид отрицательного контроля, представленный в SEQ ID NO: 36.
Другим объектом изобретения является набор для идентификации маркеров ГМИ, включающий:
а) по меньшей мере, один биочип для проведения разовой диагностики, содержащий более 6 типов дифференцирующих олигонуклеотидов, специфичных к идентифицируемым маркерам трансгенных растений, с последовательностями, представленными в SEQ ID NO: 23-33, один или более дифференцирующих олигонуклеотидов в качестве положительного контроля, с последовательностью, представленной в SEQ ID NO: 34, и один или более дифференцирующих олигонуклеотидов в качестве отрицательного контроля, с последовательностью, представленной в SEQ ID NO: 36;
б) специфические олигонуклеотиды, используемые в качестве праймеров ПЦР для идентификации маркеров ГМИ, последовательности которых представлены в SEQ ID NO: 1-22, и специфический олигонуклеотид, представленный в SEQ ID NO: 35, который комплементарен дифференцирующему олигонуклеотиду положительного контроля SEQ ID NO: 34, размещенному на биочипе в кластерах положительного контроля;
в) реагенты, которые выбирают из группы, состоящей из: реагентов для подготовки образца, реагентов для проведения мультипраймерной симметричной ПЦР, реагентов для проведения гибридизации или их комбинаций и экзонуклеазы фага лямбда.
Перечень чертежей
Фиг.1 - изображение кластеров при идентификации образца пищевого продукта, содержащего ГМИ.
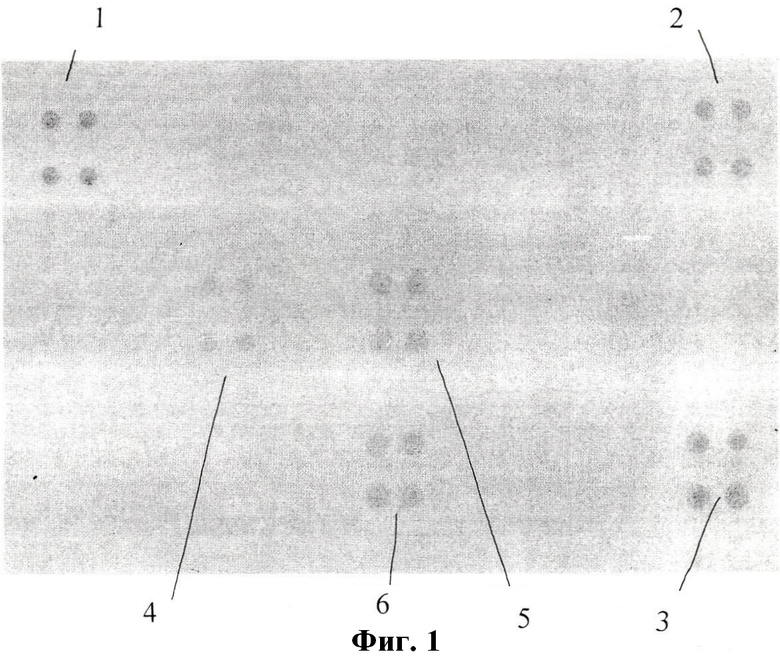
Фиг.2 - вариант схемы расположения кластеров на биочипе, где 1 - кластер отрицательного контроля, 3, 13, 15 - кластеры положительного контроля, 2 - кластер Patatin, 4 - кластер Npt II, 5 - кластер 35S, 6 - кластер Gus, 7 - кластер Lectin, 8 - кластер Nos, 9 - кластер Zein, 10 - кластер Cry1A, 11 - кластер Bar, 12 - кластер Ocs, 14 - EPSPS.
Фиг.3 - результаты гибридизации при проверке специфичности дифференцирующих праймеров, где а)-л) проверка гибридизации к дифференцирующим праймерам 35S, Nos, Ocs, NptII, Gus, Lectin, Zein, Patatin, Bar, Cry1A, EPSPS соответственно.
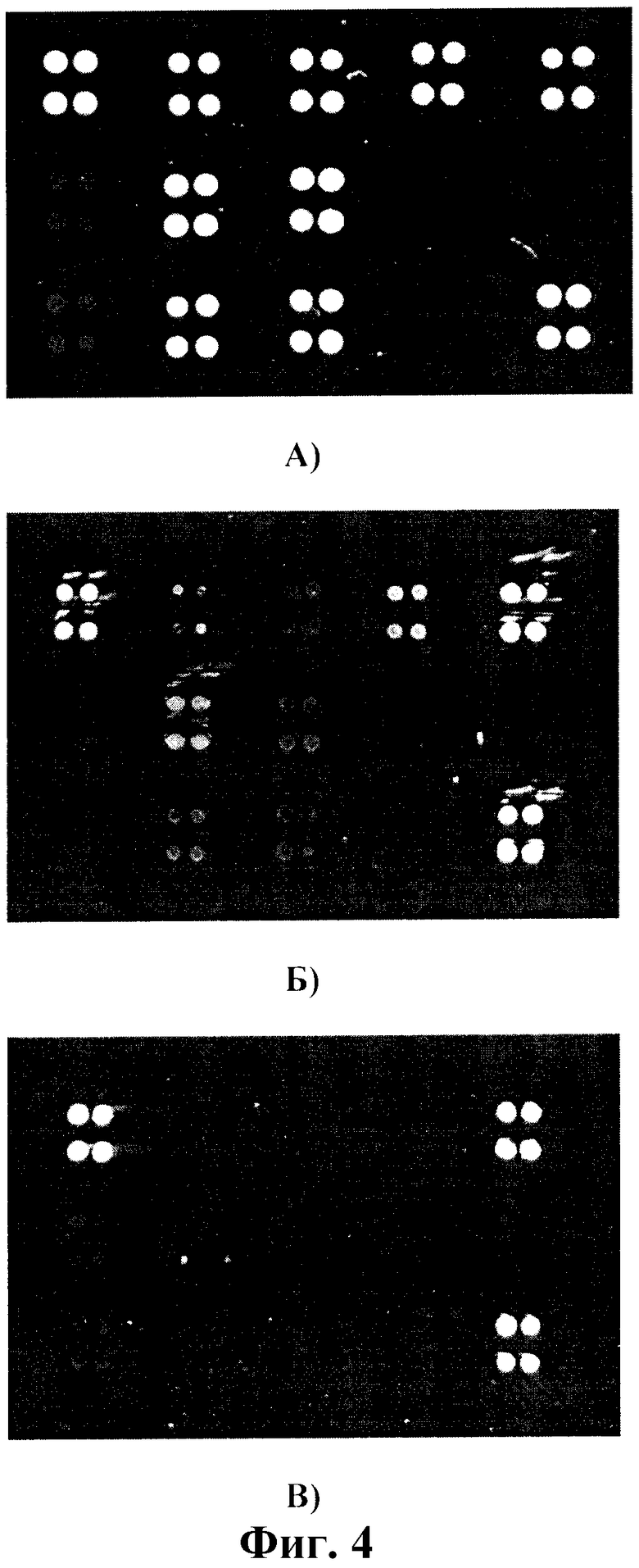
Фиг.4 - сравнение эффективности гибридизации для разных способов обработки продуктов ПЦР, где а) проба перед гибридизацией обрабатывалась экзонуклеазой, б) проба подвергалась тепловой денатурации, в) проба не обрабатывалась.
Фиг.5 - изображение кластеров, отображающих результаты мультипраймерной симметричной ПЦР и гибридизацию биочипа при выявлении маркеров: bar, cry1Ab, EPSPS, Patatin.
Описание изобретения
Способ идентификации маркеров ГМИ, в общем случае, включает следующие стадии: выбор маркеров, выбор праймеров и зондов, синтез зондов и праймеров, приготовление биочипа (например, модификация поверхности и нанесение зондов), выделение ДНК, амплификация ДНК с помощью мультипраймерной симметричной ПЦР, обработка ПЦР продукта экзонуклеазой, гибридизация ДНК на биочипе, детекция, регистрация полученного результата и интерпретация полученного результата.
Выбор маркеров
В процессе изучения способов диагностики ГМИ и известных диагностических биочипов [9, 10] было обнаружено, что большинство известных решений, связанных с выбором маркеров для диагностики, не учитывает тот факт, что в последнее время идет быстрый рост количества продуктов, содержащих генно-модифицированные растения, полученных по разным технологиям, что приводит к возможности недостоверного обнаружения продуктов питания, содержащих ГМИ.
К неочевидному решению в рамках настоящего изобретения можно отнести выбор перечня маркеров, с помощью которых осуществляют идентификацию более широкого круга трансгенных растений, чем в известных патентах аналогах. Перечень маркеров, представленный в таблице 1, составлен таким образом, что устраняются недостатки известных изобретений. В перечень, в совокупности с известными маркерами, а именно 35S промоторного участка вируса мозаики цветной капусты, гена gus из бактерии Escherichia coli, терминаторного участка гена nos из Agrobacterium tumefaciens, маркерного гена npt II из транспозона Tn5 Escherichia coli, терминаторного участка гена октопинсинтазы ocs из Agrobacterium tumefaciens, которые включены в методические указания, включены дополнительные маркеры, а именно маркер bar, представляющий ген из Streptomyces hygroscopicus, который кодирует фосфотрицин-ацетилтрансферазу; маркер cry1Ab, представляющий ген Bt-токсина насекомых из Bacillus thuringiensis, маркер EPSPS, представляющий ген 5-енолпирувилшикимат-3-фосфатсинтазы из Agrobacterium tumefaciens (CP4), обеспечивающий устойчивость к гербициду Roundup; маркер Patatin, представляющий ген (pat1) из Solanum tuberosum (картофель), который кодирует предшественник пататина; маркер Zein, представляющий ген семейства zein из Zea Mays (кукуруза); маркер Lectin, представляющий ген семейства Le1 из Glycine max (соя).
Такая совокупность маркеров позволяет реализовать техническую задачу изобретения, связанную с повышением эффективности диагностики, одновременно, быстро и с минимальными затратами диагностировать в одном анализе от 1 до 8 маркеров ГМИ, основанном на идентификации участков ДНК, специфичных для источников ГМИ и трех маркеров, относящихся к широко распространенным видам растений, таких как соя, кукуруза и картофель.
Выбор специфических и дифференцирующих олигонуклеотидов и их возможные комбинации
Выбор специфических олигонуклеотидов для ПЦР
Праймеры выбраны с учетом требований высокой специфичности к консервативным участкам генов исследуемых объектов ГМИ, что позволяет избирательно амплифицировать фрагмент ДНК трансгенного растения непосредственно из выделенного биологического образца. При выборе праймеров учитывают требования, стандартно применяемые к праймерам, в которые входят: отсутствие высокостабильных вторичных структур, малое расхождение температур плавления, не превышающее 2-3°С, а также выбор длины праймеров в диапазоне от 15 до 55 н.о.
Для составления праймеров использовались последовательности, характерные для идентифицируемого трансгенного растения и представленные в базе данных нуклеотидных последовательностей GenBank (http://ncbi.nlm.nih.gov), в которых выбирались наиболее консервативные участки, не используемые ранее и неочевидные для составления специфических и дифференцирующих олигонуклеотидов для создания биологических чипов.
Каждая пара олигонуклеотидов для ПЦР, специфичных к маркеру ГМИ, состоит из одного немеченного (не содержащего каких-либо меток, необходимых для последующего анализа и интерпретации результатов) специфического олигонуклеотида из перечисленных в списке последовательностей с любым нечетным номером с SEQ ID NO: 1 по SEQ ID NO: 21 и меченого (содержащего одну или несколько меток, необходимых для последующего анализа и интерпретации результатов) специфического олигонуклеотида из перечисленных в списке последовательностей с четным номером с SEQ ID NO: 2 по SEQ ID NO: 22, следующим сразу за первым специфическим олигонуклеотидом пары.
Выбор дифференцирующих олигонуклеотидов
Дифференцирующие олигонуклеотиды выбирали с учетом требований высокой специфичности к консервативным участкам генов исследуемых ГМИ, что позволяет проводить избирательную гибридизацию фрагментов ДНК, амплифицируемых со специфическими олигонуклеотидами при помощи ПЦР непосредственно из биологических образцов, а также исходя из отсутствия высокостабильных вторичных структур, и имеющих длину от 15 до 55 н.о., предпочтительно от 20 до 28 н.о.
При выборе дифференцирующих олигонуклеотидов одним из критериев их отбора являлось наличие гомологии только между каждым специфическим олигонуклеотидом и соответствующим маркером. Тем не менее, компьютерные методы анализа последовательностей ДНК не гарантируют возможность гибридизации дифференцирующего олигонуклеотида, специфичного к одному анализируемому маркеру, с ДНК других анализируемых маркеров, например, ввиду возможного образования ампликонами сложных вторичных структур. Поэтому для проверки специфичности дифференцирующих олигонуклеотидов был определен возможный неспецифический фон при гибридизации ДНК каждого из маркеров 35S, Nos, Ocs, Npt II, Gus, Lectin, Zein, bar, cry1Ab, EPSPS и Patatin индивидуально.
Для этого при помощи ПЦР с праймерами, специфичными только к одному из маркеров, были наработаны фрагменты ДНК каждого маркера индивидуально. Полученные пробы были обработаны экзонуклеазой и проведена гибридизация и отмывка, как указано в примере 6. Результаты гибридизации проверки специфичности дифференцирующих праймеров приведены на Фиг.3, где Фиг.3,А) для маркера 35S, Фиг.3,Б) для маркера Nos, Фиг.3,В) для маркера Ocs, Фиг.3,Г) для маркера Npt II, Фиг.3,Д) для маркера Gus, Фиг.3,Е) для маркера Lectin, Фиг.3,Ж) для маркера Zein, Фиг.3,З) для маркера Patatin, Фиг.3,И) для маркера Bar, Фиг.3,К) для маркера Cry1A, Фиг.3,Л) для маркера EPSPS.
На основании анализа результатов гибридизации была установлена максимальная величина неспецифического сигнала. С учетом этого был составлен олигонуклеотид отрицательного контроля (SEQ ID NO:36), частично комплементарный последовательности олигонуклеотида SEQ ID NO:8. Уровень сигнала при гибридизации содержащегося в ПЦР смеси флюоресцентпомеченного олигонуклеотида SEQ ID NO:8 с олигонуклеотидом SEQ ID NO:36, иммобилизованным на чипе в кластерах отрицательного контроля, достоверно превышает максимально возможный уровень сигналов «неспецифической» гибридизации и служит фоном, превышение над которым рассматривается как положительный сигнал при анализе.
Комбинации олигонуклеотидов
Представленные ниже комбинации олигонуклеотидов представляют собой наборы из двух праймеров, т.е. специфических олигонуклеотидов, и одного олигонуклеотида, используемого в качестве иммобилизованного зонда на поверхности биочипа, т.е. дифференцирующего олигонуклеотида. Для диагностики ГМИ в рамках настоящего изобретения используют следующие комбинации:
1. Для специфического выявления маркера 35S, преимущественно для идентификации 35S промотора из вируса мозаики цветной капусты (cauliflower mosaic virus), используют комбинацию, в которую входит один дифференцирующий олигонуклеотид SEQ ID NO: 23 в качестве зонда и два специфических SEQ ID NO: 1 и SEQ ID NO: 2 олигонуклеотида, используемых в качестве праймеров ПЦР.
2. Для специфического выявления маркера NOS, преимущественно для идентификации 3' нетранслируемой области гена нопалинсинтазы (NOS), входящей в Ti плазмиду Agrobacterium tumefaciens, используют комбинацию, в которую входит один дифференцирующий олигонуклеотид SEQ ID NO: 24 в качестве зонда и два специфических SEQ ID NO: 3 и SEQ ID NO: 4 олигонуклеотида, используемых в качестве праймеров ПЦР.
3. Для специфического выявления маркера OCS, преимущественно для идентификации 3' нетранслируемой области гена октопинсинтазы (OCS), входящей в Ti плазмиду Agrobacterium tumefaciens, используют комбинацию, в которую входит один дифференцирующий олигонуклеотид SEQ ID NO: 25 в качестве зонда и два специфических SEQ ID NO: 5 и SEQ ID NO: 6 олигонуклеотида, используемых в качестве праймеров ПЦР.
4. Для специфического выявления маркера NPTII, преимущественно для идентификации гена (neo, nptII) из Е.coli, кодирующего неомицин-фосфотрансферазу II, используют комбинацию, в которую входит один дифференцирующий олигонуклеотид SEQ ID NO: 26 в качестве зонда и два специфических SEQ ID NO: 7 и SEQ ID NO: 8 олигонуклеотида, используемых в качестве праймеров ПЦР.
5. Для специфического выявления маркера GUS, преимущественно для идентификации гена (uidA, GUS) из Е.coli, кодирущего бета-глюкуронидазу, используют комбинацию, в которую входит один дифференцирующий олигонуклеотид SEQ ID NO: 27 в качестве зонда и два специфических SEQ ID NO: 9 и SEQ ID NO: 10 олигонуклеотида, используемых в качестве праймеров ПЦР.
6. Для специфического выявления маркера Lectin, преимущественно для идентификации гена семейства Le1 из Glycine max (соя), который кодирует лектин, используют комбинацию, в которую входит один дифференцирующий олигонуклеотид SEQ ID NO: 28 в качестве зонда и два специфических SEQ ID NO: 11 и SEQ ID NO: 12 олигонуклеотида, используемых в качестве праймеров ПЦР.
7. Для специфического выявления маркера Zein, преимущественно для идентификации гена семейства zein из Zea Mays (кукуруза), который кодирует зеин, используют комбинацию, в которую входит один дифференцирующий олигонуклеотид SEQ ID NO: 29 в качестве зонда и два специфических SEQ ID NO: 13 и SEQ ID NO: 14 олигонуклеотида, используемых в качестве праймеров ПЦР.
8. Для специфического выявления маркера bar, преимущественно для идентификации гена из Streptomyces hygroscopicus, кодирущего фосфотрицин-ацетилтрансферазу, используют комбинацию, в которую входит один дифференцирующий олигонуклеотид SEQ ID NO: 30 в качестве зонда и два специфических SEQ ID NO: 15 и SEQ ID NO: 16 олигонуклеотида, используемых в качестве праймеров ПЦР.
9. Для специфического выявления маркера cry1Ab, преимущественно для идентификации гена из Bacillus thuringiensis, кодирующего Bt-токсин, используют комбинацию, в которую входит один дифференцирующий олигонуклеотид SEQ ID NO: 31 в качестве зонда и два специфических SEQ ID NO: 17 и SEQ ID NO: 18 олигонуклеотида, используемых в качестве праймеров ПЦР.
10. Для специфического выявления маркера EPSPS, преимущественно для идентификации гена из Agrobacterium tumefaciens (CP4), кодирующего 5-енолпирувилшикимат-3-фосфатсинтазу, используют комбинацию, в которую входит один дифференцирующий олигонуклеотид SEQ ID NO: 32 в качестве зонда и два специфических SEQ ID NO: 19 и SEQ ID NO: 20 олигонуклеотида, используемых в качестве праймеров ПЦР.
11. Для специфического выявления маркера Patatin, преимущественно для идентификации гена pat1 из Solanum tuberosum (картофеля), кодирующего предшественник пататина, используют комбинацию, в которую входит один дифференцирующий олигонуклеотид SEQ ID NO: 33 в качестве зонда и два специфических SEQ ID NO: 21 и SEQ ID NO: 22 олигонуклеотида, используемых в качестве праймеров ПЦР.
Ниже приведен перечень специфических нуклеотидов (таблица 2) и перечень дифференцирующих олигонуклеотидов (таблица 3), используемых в рамках настоящего изобретения.
Синтез зондов и праймеров
Олигонуклеотиды синтезируют на автоматическом синтезаторе (например, модель "Gene Assembler" фирмы Pharmacia) стандартным амидофосфитным методом. Дифференцирующие олигонуклеотиды содержат 5'- или 3'-концевую группу, обеспечивающую их иммобилизацию на поверхности чипа, например, 5'-концевую фосфатную группу, которую вводят при помощи химического фосфорилирования в процессе синтеза. Один из пары специфических олигонуклеотидов для проведения ПЦР модифицируют по 5'-концу меткой, которую выбирают из группы, состоящей из: каталитической, лигандной, флуоресцентной и радиоактивной, которую вводят химическими методами или энзиматически.
Выделение ДНК и подготовка материала для амплификации
Выделение ДНК проводят из биологического образца, содержащего ГМИ, входящего в группу, состоящую из плодов или сока растений, зерна, муки или пищевых продуктов, в состав которых входят генетически модифицированные растения.
Выделение ДНК проводят с использованием общепринятых методик, основанных, например, на щелочном лизисе, использовании детергентов, протеиназы К, экстракции фенолом, хлороформом и/или смесью фенол/хлороформ, либо очистку на диатомовой земле или с помощью диоксида кремния.
Положительные и отрицательные контроли
В рамках данного изобретения положительный контроль используется в биочипе и в диагностическом наборе для подтверждения соблюдения оптимальных условий проведения процедур, необходимых для идентификации заявленных трансгенных растений, а именно гибридизации и детекции. С этой целью на чип нанесен дифференцирующий олигонуклеотид SEQ ID NO: 34, которому комплементарен олигонуклеотид SEQ ID NO: 35. Олигонуклеотид положительного контроля добавляют к пробе после ПЦР или обработки экзонуклеазой фага лямбда и проводят гибридизацию в тех же условиях, что и для определяемых маркеров.
Отрицательные контроли в рамках данного изобретения используют для подтверждения соблюдения оптимальных условий гибридизации. В качестве отрицательного контроля на чип иммобилизуют олигонуклеотид, частично комплементарный последовательности ДНК положительного контроля, или олигонуклеотид, частично комплементарный какому-либо из специфических праймеров для ПЦР, которые модифицированы по 5'-концу меткой из группы, приведенной в п. «Синтез зондов и праймеров». В последовательности дифференцирующего олигонуклеотида, служащего в качестве отрицательного контроля, количество и положение нуклеотидов, несоответствующих последовательности ДНК положительного контроля или специфическим олигонуклеотидам для ПЦР, подбирают таким образом, чтобы этот дифференцирующий олигонуклеотид был способен образовывать и сохранять несовершенный дуплекс с одноцепочечной ДНК положительного контроля или специфического олигонуклеотида для ПЦР при несоблюдении условий гибридизации и отмывки, а именно при проведении гибридизации и отмывки в условиях, более мягких по сравнению с оптимальными. В качестве примера, который включает, но не ограничивает объем изобретения, в перечне последовательности приведен дифференцирующий олигонуклеотид ID SEQ NO: 36, который иммобилизуют на поверхность чипа и используют в качестве отрицательного контроля.
Выбор метки
Известно большое число патентов, в которых рассматриваются разные типы меток для построения биологических чипов. Известна классификация меток на группы, состоящие из: каталитических, лигандных, флуоресцеитных или радиоактивных молекул [9]. Указанные патенты приведены в качестве ссылок. Каталитическую молекулу выбирают из группы, включающей гемин, цианкобаламин или флавин. Лигандную молекулу выбирают из группы, включающей биотин, диоксигенин или динитробензол. Флуоресцентную молекулу выбирают из группы, включающей FAM, TAMRA, Cy3, Cy5, Cy7, R6G, R110, ROX или JOE.
Амплификация ДНК при помощи ПЦР
Однонитевые фрагменты ДНК типичных последовательностей ГМИ, приведенных в таблице 1, получают в реакции симметричной ПЦР с использованием высокоспецифичных праймеров.
Условия ПЦР
В состав ПЦР-смеси входит 1x-кратный реакционный буфер, например, от 10 до 80 мМ трис-HCl рН 8,8, до 50 мМ KCl и/или до 25 mM (NH4)2SO4, и/или 0,1% тритон Х-100, и/или 0,1% Tween 20, и/или 0,8% Nonidet P40, от 1 мМ до 5 мМ, оптимально от 1,5 мМ до 2,5 мМ, MgCl2 или MgSO4, от 10 мкг до 300 мкг, оптимально от 50 мкг до 200 мкг, ДНК анализируемого образца и не менее одной пары специфических олигонуклеотидных праймеров для каждого маркера. Общее количество пар специфических олигонуклеотидов может варьировать от 1 до 11 в сочетаниях для идентификации, по меньшей мере, одного маркера ГМИ. В состав ПЦР-смеси входит от 1 ед. до 20 ед., оптимально от 3 ед. до 7 ед., наиболее оптимально от 4 ед. до 6 ед. активности Taq-полимеразы. В состав ПЦР смеси могут входить реагенты, повышающие специфичность и эффективность ПЦР, например, такие как бетаин, эктаин и его производные, а также трегалоза, диметилсульфоксид, глицерин.
Концентрация олигонуклеотидов в ПЦР может составлять от 0,05 мкМ до 0,3 мкМ, оптимально от 0,1 мкМ до 0,25 мкМ.
Объем реакционной смеси может составлять от 5 мкл до 200 мкл, оптимально от 20 мкл до 50 мкл. Реакцию можно проводить под минеральным маслом или без него, в зависимости от конструкции ДНК-амплификатора. ПЦР проводят с использованием ДНК амплификатора (например, «Терцик», ДНК-технология, Россия или «Mastercycler», Eppendorf, Германия) в условиях, приведенных в таблице 4.
Обработка ПЦР продуктов экзонуклеазой фага лямбда
Один из пары праймеров для ПЦР содержит фосфатную группу на 5'-конце. Это необходимо для того, чтобы цепь фрагмента ДНК, которая синтезируется в ходе ПЦР с этим праймером, эффективно гидролизовалась экзонуклеазой фага лямбда. Поскольку второй из пары праймеров на 5'-конце содержит биотин или флуоресцентную метку, эта цепь ДНК защищена от деградации экзонуклеазой. Это позволяет после обработки продуктов ПЦР экзонуклеазой фага лямбда получать одноцепочечную ДНК, меченную на 5'-конце биотином или флуоресцентной меткой. Гибридизация с одноцепочечной ДНК проходит более эффективно, что позволяет повысить чувствительность и воспроизводимость диагностики. Для получения одноцепочечной ДНК часто используют асимметричную ПЦР. Метод получения одноцепочечной ДНК с помощью асимметричной ПЦР требует большого количества циклов ПЦР и до 50-кратного избытка одного из праймеров, что приводит к увеличению времени проведения ПЦР и увеличивает расходы на проведение реакции. Применение экзонуклеазы фага лямбда для получения одпоцепочечной ДНК позволяет использовать стандартную симметричную ПЦР, которая более эффективна и экономична как по количеству реагентов, так и по затрачиваемому времени. Для проведения гидролиза продуктов ПЦР используют от 5 до 20 единиц активности экзонуклеазы фага лямбда. Время обработки составляет от 30 до 90 мин. Более предпочтительно использовать 10 единиц фермента и проводить реакцию 60 минут при температуре 37°С.
Проведение гибридизации
Меченые продукты ПЦР гибридизуются с ДНК-зондами (дифференцирующими олигонуклеотидами), иммобилизованными на биочипе, в специально подобранном буфере.
Условия гибридизации
Гибридизацию проводят в буфере, содержащем 0,5×-5×SSC, 0,1% SDS, до 25% формамида и/или до 10 мМ ЭДТА от 30 мин до 1 часа при 35-50°С. Затем чип промывают при комнатной температуре на орбитальном шейкере от 5 до 10 раз растворами 0,5×-5×SSC, 0,1% SDS. В случае использования ферментов для проявления результатов гибридизации последние три промывки проводят раствором SSC, не содержащим SDS.
Проведение пероксидазной реакции
В случае, если продукты ПЦР мечены биотипом, для дальнейшей идентификации необходимо провести реакцию с конъюгатом стрептавидин-пероксидаза или стрептавидин-фосфатаза.
Структура биочипа
Биочип для идентификации ГМИ представляет собой несущий элемент, на поверхности которого иммобилизованы более 6 кластеров с дифференцирующими олигонуклеотидами, выбранными из SEQ ID NO: 23-33, по меньшей мере, один дифференцирующий олигонуклеотид положительного контроля, представленный в SEQ ID NO: 34, и, по меньшей мере, один олигонуклеотид отрицательного контроля, представленный в SEQ ID NO: 36. Дифференцирующие олигонуклеотиды нанесены в кластере в четырех или девяти повторах. Количество кластеров, размещенных в рабочей зоне каждого индивидуального чипа, может составлять 15. На Фиг.2 приведен один из вариантов структурной схемы биочипа, который включает, но не ограничивает возможности использования других вариантов, например, представленный на Фиг.1 с тремя положительными контролями.
Предпочтительной формой мультичипа является плоский прямоугольный элемент, выполненный из твердого или гибкого материала разной толщины. Толщина несущего элемента может составлять от 0,05 мм до 5 мм. Несущий элемент чипа выполнен из материалов, входящих в группу, состоящую из: полимеров, стекла, металлов, керамики или их комбинаций. Полимеры выбирают из группы, состоящей из: полиметилметакрилата, полибутилметакрилата, поливинилхлорида, поликарбоната, сополимеров метилметакрилата и/или сополимеров бутилметакрилата с другими мономерами, такими как стирол, акрилонитрил.
Поверхность несущего элемента может быть модифицирована нужным образом или покрыта каким-либо материалом, способным иммобилизовать олигонуклеотиды.
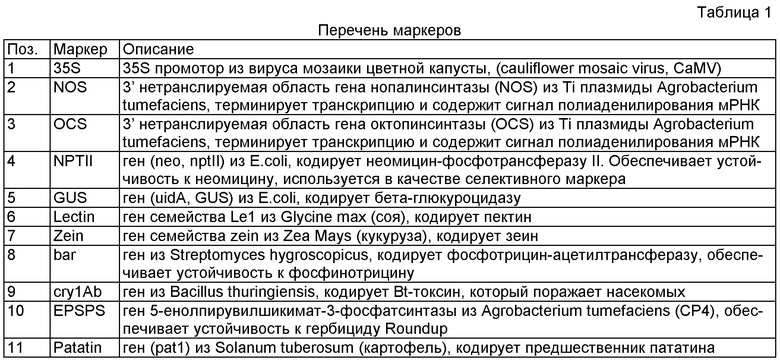
В одном из вариантов биочипа, который включает, но не ограничивает объем изобретения, расположение кластеров положительного контроля выполнено таким образом, что кластеры положительного контроля располагают, по меньшей мере, в двух диагонально расположенных углах рабочей зоны, обозначая таким образом границы рабочей зоны, что упрощает процедуру идентификации расположения рабочей зоны при сканировании результатов диагностики. На фиг.1 приведена гибридизационная картина, полученная при сканировании биочипа при диагностике пищевого продукта, в которой приведен пример размещения трех положительных контролей в углах рабочей зоны биочипа (раздел «интерпретация данных»).
Идентификация мультичипа
Идентификатор биочипа может включать в себя код номера партии и/или даты изготовления, информацию о перечне размещенных дифференцирующих олигонуклеотидов. Идентификатор может быть выполнен в виде штрих-кода или элемента с магнитной записью информации.
Обращение к уникальному коду биочипа обеспечит быстрый поиск информации в базах данных о всех образцах при параллельном скрининге ГМИ.
Интерпретация данных анализа
Интенсивность сигнала образующихся совершенных или несовершенных дуплексов внутри каждой группы кластеров существенно выше интенсивности сигнала отрицательных контролей, что позволяет проводить надежную идентификацию ГМИ. Детектирование и/или идентификацию ГМИ осуществляют путем сравнительного анализа уровня сигналов исследуемого образца, положительного и отрицательного контролей. Интерпретацию результатов проводят исходя из гибридизационной картины, которую получают используя экспериментальную установку, включающую, например, сканер, компьютер и специальное программное обеспечение. На фиг.1 приведена гибридизационная картина с колориметрическими метками, полученная при сканировании биочипа, для определения возможного содержания ГМИ в пищевой пробе, выделенной из 0.1% сои.
В данном варианте биочипа, по трем углам рабочей зоны биочипа выявлены кластеры 1, 2, 3 положительного контроля, имеющие максимальный сигнал. Во втором горизонтальном ряду расположен кластер 4, соответствующий маркеру S35, и кластер 5, соответствующий маркеру Nos. В третьем горизонтальном ряду расположен кластер 6, соответствующий маркеру Lectin.
Интерпретацию результатов проводят следующим образом. Интенсивности флуоресцентных или колориметрических сигналов в каждой группе ДНК-зондов, в которой содержатся олигонуклеотиды, видоспецифичные для одного из 11 маркеров ГМИ, с помощью специального программного обеспечения сравнивают с группами зондов положительного (максимальная интенсивность сигнала) и отрицательного (минимальная интенсивность сигнала) контроля. В результате образуется гибридизационная картина, различная для групп положительного контроля, отрицательного контроля и для кластеров, характеризующих маркеры ГМИ, что позволяет однозначно интерпретировать полученные данные.
Набор для диагностики
Другим объектом изобретения является набор для идентификации маркеров ГМИ, включающий:
а) по меньшей мере, один биочип для проведения разовой диагностики, содержащий более 6 дифференцирующих олигонуклеотидов, специфичных к идентифицируемым маркерам трансгенных растений, с последовательностями представленными в SEQ ID NO: 23-33, один или более дифференцирующих олигонуклеотидов в качестве положительного контроля, с последовательностью, представленной в SEQ ID NO: 34, и один или более дифференцирующих олигонуклеотидов в качестве отрицательного контроля, с последовательностью, представленной в SEQ ID NO: 36;
б) специфические олигонуклеотиды, используемые в качестве праймеров ПЦР для идентификации маркеров ГМИ, последовательности которых представлены в SEQ ID NO: 1-22, и специфический олигонуклеотид, представленный в SEQ ID NO: 35, который комплементарен дифференцирующему олигонуклеотиду положительного контроля SEQ ID NO: 34, размещенному на биочипе в кластерах положительного контроля;
в) реагенты, которые выбирают из группы, состоящей из: реагентов для подготовки образца, реагентов для проведения мультипраймерной симметричной ПЦР, реагентов для проведения гибридизации или их комбинаций и экзонуклеазы фага лямбда.
Примеры
Ниже приведены примеры, которые включают, но не ограничивают объем изобретения.
Пример 1. Приготовление поверхности биочипа (модификация поверхности)
Матрицу, содержащую аминогруппы с плотностью 1-2 групп/нм2, изготавливают с применением раствора 3-аминопропилтриэтоксисилана (АПТЭС) в различных растворителях в зависимости от типа подложек. Например, стеклянные подложки инкубировали в течение часа в растворе 3-аминопропилтриэтоксисилана в ацетоне или в этаноле. Концентрацию АПТЭС варьировали от 0,1 до 10% по объему. Если реакцию проводят в ацетоне, стекла трижды промывали ацетоном по 5 минут, затем этанолом 2 раза по 3 минуты и сушили при температуре 120-130°С в течение 20 минут.
Пример 2. Пришивка олигонуклеотидных зондов к поверхности амино-модифицированного стекла
Нанесение зондов
Реакционную смесь, содержащую 1 мкМ олигонуклеотида и 60 мМ гидрохлорида 1-этил-3-(3-диметиламинопропил)-карбодиимида в 0,1 М 1-метилимидазоле, рН 7, наносили на поверхность матрицы роботом (Xpress Lane, США). Объем капель каждого зонда составляет не менее 0,005 мкл. Матрицы с нанесенными олигонуклеотидами помещали в герметичную емкость, выдерживали при комнатной температуре в течение 1 часа во влажной атмосфере и промывали на шейкере 0,5 мМ раствором NaCl, в содержащим 0,1% SDS, в течение 10 минут, затем дистиллированной водой. Биочип высушивали и хранили при температуре от 4 до 8°С. Концентрация олигонуклеотида в объеме капель каждого зонда составляет от 0,5 до 100 мкмоль/л.
В качестве зондов использовали дифференцирующие олигонуклеотиды, выбираемые из ID SEQ No: 23-33. Олигонуклеотиды наносили кластерами по 4 точки.
Пример 3. Проведение мультипраймерной симметричной ПЦР для гибридизации биочипа с семью кластерами для выявления маркеров Zein, Lectin, NOS, 35S, OCS, NPTII, GUS
ПЦР-смесь состояла из 10 мМ трис-HCl рН 8,8, 50 мМ KCl, 0,8% Nonidet P40, 2 мМ MgCl2, 100 нг ДНК анализируемого образца, специфические олигонуклеотидные праймеры ID SEQ NO: 1, 3, 5, 7, 9, 11, 13 и праймеры ID SEQ NO: 2, 4, 6, 8, 10, 12, 14, модифицированные биотином по 5'-положению, попарно используют в концентрациях: ID SEQ NO: 1 и ID SEQ NO: 2 в концентрации 0,25 мкМ; ID SEQ NO: 3 и ID SEQ NO: 4 в концентрации 0,25 мкМ; ID SEQ NO: 5 и ID SEQ NO: 6 в концентрации 0,1 мкМ; ID SEQ NO: 7 и ID SEQ NO: 8 в концентрации 0,1 мкМ; ID SEQ NO: 9 и ID SEQ NO: 10 в концентрации 0,1 мкМ; ID SEQ NO: 11 и ID SEQ NO: 12 в концентрации 0,15 мкМ; ID SEQ NO: 13 и ID SEQ NO: 14 в концентрации 0,15 мкМ, и 5 ед. Taq-полимеразы в объеме 20 мкл. Реакцию проводили на ДНК-амплификаторе Mastercycler (Eppendorf, Германия) при условиях, приведенных в таблице 5.
Пример 4. Проведение мультипраймерной симметричной ПЦР для гибридизации биочипа и выявления маркеров: bar, cry1Ab, EPSPS, Patatin
ПЦР-смесь состояла из 10 мМ трис-HCl рН 8,8, 50 мМ KCl, 0,8% Nonidet P40, 2 мМ MgCl2, 100 нг ДНК анализируемого образца, специфические олигонуклеотидные праймеры ID SEQ NO: 15, 17, 19, 21 и праймеры ID SEQ NO: 16, 18, 20, 22. Олигонуклеотиды, представленные в SEQ ID NO: 15, 17, 19, 21, содержали 5'-концевой фосфат. Олигонуклеотиды, представленные в SEQ ID NO: 16, 18, 20, 22, содержали на 5'-конце флюоресцентную метку Cy5. Праймеры попарно используют в концентрациях: ID SEQ NO: 15 и ID SEQ NO: 16 в концентрации 0,25 мкМ; ID SEQ NO: 17 и ID SEQ NO: 18 в концентрации 0,25 мкМ; ID SEQ NO: 19 и ID SEQ NO: 20 в концентрации 0,25 мкМ; ID SEQ NO: 21 и ID SEQ NO: 22 в концентрации 0,25 мкМ и 5 ед. Taq-полимеразы в объеме 20 мкл. Реакцию проводили на ДНК-амплификаторе Mastercycler (Eppendorf, Германия) при условиях, приведенных в таблице 5.
Пример 5. Гибридизация
К 20 мкл ПЦР смеси добавляли 5 мкл 20х SSC и 2,5 мкл 10% SDS, наносили на рабочую зону подложки, накрывали пленкой размером 16×21 мм и инкубировали в течение 1 часа при 45°С. По окончании реакции гибридизации поверхность чипа промывали на орбитальном шейкере следующими растворами:
A) 2× SSC, 0,1% SDS - 2 раза по 5 минут;
Б) 0,2× SSC, 0,1% SDS - 2 раза по 5 минут;
B) 0,2× SSC - 3 раза по 1 минуте.
По окончании промывки биочип высушивали при комнатной температуре.
Пример 6. Проведение пероксидазной реакции
Исходный стрептавидин-пероксидазный конъюгат ("Имбио", Россия) разбавляли в 200 раз 1x SSC, содержащим 1% БСА. Конъюгат наносили на рабочую зону подложки, накрывали пленкой и выдерживали во влажной атмосфере при комнатной температуре в течение 30 минут. Отмывку несвязавшегося конъюгата проводили 2х SSC в течение 5 минут, 1x SSC - 5 минут и 0,1×SSC в течение 1 минуты.
Подложку заливали свежеприготовленным раствором субстрата - диметиламинобензидина. Для этого, не более чем за полчаса до проведения реакции, растворяли 1 мг DAB в 1 мл 0,1×SSC, добавляли 20 мкл 3%-ного раствора пероксида водорода и перемешивали. Рабочую зону заливали приготовленным раствором субстрата, накрывали пленкой и выдерживали от 10 до 30 минут при комнатной температуре. В случае положительной реакции появлялись коричневые пятна окисленного субстрата. Затем стекло промывали дистиллированной водой и помещали в вертикальном положении в контейнер для осушки и дальнейшего хранения.
Пример 7. Детекция зондов
Для получения изображений гибридизационных микрозон использовали слайд сканер Nikon CoolScan 9000ED. Затем проводили количественную обработку изображений с помощью компьютерной программы.
На фиг.1 представлена картина гибридизации образцов ДНК. Видно, что для каждой выявленной ДНК характерна своя индивидуальная гибридизационная картина. Кластеры, соответствующие отрицательному контролю, не проявились. Положительные контроли расположены в трех углах рабочей зоны биочипа и определяют границы рабочей зоны при количественной обработке изображения.
Для получения изображений кластеров с использованием флюоресцентных меток использовали флюоресцентный чип-детектор, изготовляемый фирмой ЗАО «Молекулярно-медицинские технологии». На фиг.5 представлено изображение результата мультипраймерной симметричной ПЦР и гибридизации биочипа при выявлении маркеров: bar, cry1Ab, EPSPS, Patatin.
Пример 8. Проверка эффективности гибридизации после обработки продуктов ПЦР препаратом экзонуклеазы фага лямбда
Получили 3 идентичные ПЦР-смеси в объеме 20 мкл каждая. Состав ПЦР смеси: 67 мМ Трис-HCl, рН 8.8; 16 мМ (NH4)2SO4; 2 мМ MgCl2; 0,2 мМ дНТФ; 0,01% Твин 20, по 0,25 мкМ каждого олигоиуклеотида, которые представленны в SEQ ID №1-14. Олигонуклеотиды, представленные в SEQ ID №1, 3, 5, 7, 9, 11 и 13, содержали 5'-концевой фосфат. Олигонуклеотиды, представленные в SEQ ID №2, 4, 6, 8, 10, 12 и 14, содержали на 5'-конце флюоресцентную метку Cy5, смесь рекомбинантных плазмидных ДНК, содержащих клонированные фрагменты маркеров 35S, NOS, OCS, NPTII, GUS, LECTIN и ZEIN, в количестве 0,05 нг каждая и 5 ед. активности Taq-полимеразы. Условия ПЦР приведены в таблице 6.
После ПЦР к пробе 1 добавили 1 мкл препарата экзонуклеазы фага лямбда, содержащего 10 единиц активности, и инкубировали 1 час при 37°С. Реакцию останавливали добавлением 2 мкл стоп-раствора (состав стоп-раствора: 100 мМ ЭДТА и 0,5 мкМ олигопуклеотида, представленного в SEQ ID NO: 35, с флюоресцентной меткой Cy5 на 5'-конце) и инкубировали пробу 10 мин при 80°С. К пробам 2 и 3 был добавлен стоп-раствор. Затем пробу 2 для денатурации ДНК инкубировали в кипящей бане в течение 5 мин, а затем помещали в лед. Проба 3 перед гибридизацией не подвергалась никаким обработкам, т.е. служила в качестве контроля эффективности использования экзонуклеазы и тепловой денатурации ДНК.
После этого проводили гибридизацию. Для этого в каждую микропробирку с пробами 1, 2 и 3 добавили 2,5 мкл 20× SSC и 2,5 мкл 1% SDS и перемешали пилотированием. Полученную смесь нанесли на середину рабочей зоны микрочипа (для каждой пробы использовали отдельный микрочип), накрыли пленкой размером 16×21 мм, поместили микрочипы во влажную камеру и инкубировали 1 час при температуре 45°С в воздушном термостате. По окончании реакции удалили пленку с каждого микрочипа и провели отмывку на орбитальном шейкере следующими растворами (температура растворов составляла 28°С):
По окончании промывки микрочипы подсушивали при комнатной температуре в течение 15 мин и проводили детекцию результатов при помощи чип-детектора «ВАФ-1». Результаты приведены на Фиг.4: а) - для пробы 1, б) - для пробы 2 и в) - для пробы 3. Наибольшая интенсивность сигнала в кластерах анализируемых генетических маркеров наблюдалась после обработки пробы 1 экзонуклеазой. Для пробы 2, подвергавшейся тепловой денатурации, не наблюдалось максимального насыщения флюоресценции, тогда как в контрольной пробе 3 положительный сигнал на гибридизацию наблюдался только в кластерах положительных и отрицательных контролей. Данный пример демонстрирует, что использование экзонуклеазы существенно увеличивает эффективность анализа.
Промышленная применимость
Способ идентификации ГМИ на биочипе выгодно отличается быстротой проведения анализа, простотой используемой методики подготовки образца ДНК для гибридизации, возможностью одновременного выявления до 11 наиболее широко распространенных маркеров ГМИ, возможностью проведения анализа в лабораторных или полевых условиях, а также относительной дешевизной. Тексты патентов и патентных заявок рассматриваются в тексте данного описания в качестве ссылок.
Литература
1. Липп М.И др. Постановление Европейского Парламента и Совета №1829/2003 от 22.09.2003 г. "О генетически модифицированной пище и кормах". Журнал АОАС Интерн., том.82, №4, рр.923-928 (1999).
2. Park H. et al. Detection method of genetic recombinant food and detection kit of that. WO 2001098523 (27.12.2001).
3. Lauter F.R. et al. Test kit and method for quantitatively detecting genetically modified DNA in foodstuff by means of fluorescence-coupled PCR. US Patent Apll. 20030148278 (August 7, 2003).
4. Hurst М. et al. Method, apparatus and system for quantifying the content of genetically modified material in a sample. US Patent Apll. 20070015186 (January 18, 2007).
5. Changhai Y. et al. DNA extracting method and use thereof in detecting transgene component in food. CN 1420124 (2003-05-28).
6. Ye С. et al. Method for detecting transgenic plant and products thereof. CN 1385541 (2002-12-18).
7. Kim J. et al. Method and kit for detection genetically modified organism by polymerase chain reaction (PCR). KR 20020066413 (2002-08-17).
8. Национальный стандарт Российской Федерации ГОСТ Р 52173-2003 «Сырье и продукты пищевые. Метод идентификации генетически модифицированных источников (ГМИ) растительного происхождения».
9. Белецкий И.П. и др. Набор праймеров для детекции и/или идентификации трансгенных последовательностей ДНК в растительном материале и его содержащих продуктах (варианты), праймер (варианты), пара праймеров (варианты), способ детекции и/или идентификации с их использованием (варианты) и устройство для осуществления способа. Патент RU 2265668 (2005.12.10).
10. Мирзабеков А.Д. и др. Способ идентификации трансгенных последовательностей ДНК в растительном материале и продуктах на его основе, набор олигонуклеотидов и биочип для осуществления этого способа. Патент RU 2270254 (2006.02.20).
11. Remacle J. et al. Real-time PCR of targets on a micro-array. WO 2006053770 (2006-05-26).
12. Национальный стандарт Российской Федерации ГОСТ Р 52174-2003 Продукты пищевые. Биологическая безопасность. Генетически модифицированные организмы и источники. Метод идентификации генетически модифицированных растений и продуктов на их основе.
13. Kang J. et al. Detection primer for genetically modified organism and manufactured goods and detection kit using same. KR 20030018218 (2003-03-06).
14. Akiyama H. et al. Method for quantitatively detecting recombinant proceseed food. JP 2006314237 (2006-11-24).
15. Gong Y. et al. Transgenic agricultural product detection kit. CN 1428438 (2003-07-09).
16. Методические указания МУК 4.2.2008-05 «Метод идентификации генно-инженерно-модифицированных организмов (ГМО) растительного происхождения с применением ферментного анализа на биологическом микрочипе».
17. Анкенбауер В.И. и др. Способ амплификации ДНК и композиция для его осуществления. Патент RU 2260055 (2005.09.10).
18. Nikiforov T. et al. Method for generating single-stranded DNA molecules. US Pat. 5,518,900 May 21, 1996.
19. Rudi К. et al. Methods of nucleic acid amplification. US Pat. Applic. 20060035222 (February 16, 2006).
20. Референсные материалы ГМИ. Материалы фирмы ЗАО "БиоХимМак" (http://www.biochemmack.ru/product/diagnostics/moleculardiagnostics/GMI/).
| название | год | авторы | номер документа |
|---|---|---|---|
| ДИФФЕРЕНЦИРУЮЩИЙ И СПЕЦИФИЧЕСКИЙ ОЛИГОНУКЛЕОТИДЫ ДЛЯ ИДЕНТИФИКАЦИИ ПОСЛЕДОВАТЕЛЬНОСТЕЙ ДНК ИНФЕКЦИОННЫХ АГЕНТОВ В БИОЛОГИЧЕСКИХ МАТЕРИАЛАХ, СПОСОБ ВИДОВОЙ ИДЕНТИФИКАЦИИ ИНФЕКЦИОННЫХ АГЕНТОВ, БИОЧИП И НАБОР ДЛЯ ОСУЩЕСТВЛЕНИЯ ЭТОГО СПОСОБА | 2006 |
|
RU2348695C2 |
| НАБОР ДИФФЕРЕНЦИРУЮЩИХ И СПЕЦИФИЧЕСКИХ ОЛИГОНУКЛЕОТИДОВ ДЛЯ ИДЕНТИФИКАЦИИ ДНК ВОЗБУДИТЕЛЕЙ ОСТРЫХ КИШЕЧНЫХ ИНФЕКЦИЙ, СПОСОБ ИДЕНТИФИКАЦИИ ОКИ, МИКРОЧИП И ДИАГНОСТИЧЕСКАЯ СИСТЕМА ДЛЯ ОСУЩЕСТВЛЕНИЯ СПОСОБА | 2010 |
|
RU2509804C2 |
| СПОСОБ ИЗГОТОВЛЕНИЯ МНОГОФУНКЦИОНАЛЬНОГО МУЛЬТИЧИПА, МУЛЬТИЧИП ДЛЯ ПОСЛЕДОВАТЕЛЬНОГО ИЛИ ПАРАЛЛЕЛЬНОГО СКРИНИНГА БИОПОЛИМЕРОВ, СПОСОБ АНАЛИЗА БИОПОЛИМЕРОВ И НАБОР ДЛЯ ОСУЩЕСТВЛЕНИЯ СПОСОБА | 2006 |
|
RU2321638C2 |
| БИОЧИП С МНОГОФУНКЦИОНАЛЬНОЙ СИСТЕМОЙ ИДЕНТИФИКАЦИИ (ВАРИАНТЫ) | 2007 |
|
RU2386970C2 |
| ДИАГНОСТИЧЕСКИЙ ТЕСТ И НАБОР ДЛЯ ДЕТЕКТИРОВАНИЯ БИОМОЛЕКУЛ | 2007 |
|
RU2424322C2 |
| ТРАНСФОРМИРУЕМЫЙ БИОЧИП | 2007 |
|
RU2436843C2 |
| НАБОР ПРАЙМЕРОВ ДЛЯ ДЕТЕКЦИИ И/ИЛИ ИДЕНТИФИКАЦИИ ТРАНСГЕННЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ ДНК В РАСТИТЕЛЬНОМ МАТЕРИАЛЕ И ЕГО СОДЕРЖАЩИХ ПРОДУКТАХ (ВАРИАНТЫ), ПРАЙМЕР (ВАРИАНТЫ), ПАРА ПРАЙМЕРОВ (ВАРИАНТЫ), СПОСОБ ДЕТЕКЦИИ И/ИЛИ ИДЕНТИФИКАЦИИ С ИХ ИСПОЛЬЗОВАНИЕМ (ВАРИАНТЫ) И УСТРОЙСТВО ДЛЯ ОСУЩЕСТВЛЕНИЯ СПОСОБА | 2004 |
|
RU2265668C1 |
| НАБОР ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ ДЛЯ ИДЕНТИФИКАЦИИ ДНК ЖИВОТНЫХ В КОРМАХ И МЯСНЫХ ПРОДУКТАХ | 2011 |
|
RU2560579C2 |
| ВЫСОКОСПЕЦИФИЧНЫЙ ДНК-МАРКЕР, ИСПОЛЬЗУЕМЫЙ В КАЧЕСТВЕ ЭНДОГЕННОГО РЕФЕРЕНСНОГО КОНТРОЛЯ ДЛЯ ОБНАРУЖЕНИЯ ГЕНОМНОЙ ДНК КАРТОФЕЛЯ В РАСТИТЕЛЬНОМ МАТЕРИАЛЕ И ПИЩЕВЫХ ПРОДУКТАХ, В ТОМ ЧИСЛЕ ПРИ ИДЕНТИФИКАЦИИ ГМО | 2013 |
|
RU2539756C1 |
| СПОСОБ ПОЛУЧЕНИЯ ДНК-ЧИПОВ | 2006 |
|
RU2321574C2 |


Изобретение относится к области генной инженерии и предназначено для выявления типичных последовательностей ДНК различных трансгенных растений. Способ идентификации последовательностей ДНК в биологическом материале включает выделение нуклеиновых кислот, проведение мультипраймерной симметричной полимеразной цепной реакции с использованием выделенных из анализируемого материала ДНК и последующую гибридизацию флуоресцентно-меченных или биотинилированных продуктов этой реакции на специализированных биочипах. Предложены набор олигонуклеотидов, биологический микрочип, содержащий до 11 типов дифференцирующих олигонуклеотидов и набор реактивов для проведения такого способа, также варианты составов комбинаций олигонуклеотидов для реализации способа. Изобретение позволяет ускорить проведение анализа на наличие ГМИ в пищевых продуктах и повысить его точность. 10 н. и 8 з.п. ф-лы, 5 ил., 6 табл., 8 пр.
1. Способ идентификации маркеров генетически модифицированных растений, включающий анализ образца с исследуемыми ДНК с использованием прямых и обратных праймеров, специфичных к генам маркеров ГМИ (генетически модифицированных источников), и биочипа, проведение мультипраймерной ПЦР в образце, обработку продуктов ПЦР реакции ферментом, инкубацию ПЦР продукта с дифференцирующими олигонуклеотидами, иммобилизированными на поверхности биочипа в условиях гибридизации, определение наличия маркеров ГМИ детекцией образованных гибридизационных комплексов на биочипе, отличающийся тем, что идентифицируют маркеры ГМИ, перечисленные в табл.1, биочип содержит более 6 типов кластеров дифференцирующих олигонуклеотидов, выбираемых из последовательностей, представленных в SEQ ID NO: 23-33, при этом в качестве специфичных праймеров используют праймеры, последовательности которых выбирают из группы, состоящей из SEQ ID NO: 1-22, в качестве ПЦР используют мультипраймерную симметричную ПЦР, продукты которой предпочтительно в объеме 20 мкл обрабатывают предпочтительно 1 мкл раствора экзонуклеазы фага лямбда в концентрации от 5 до 20 единиц активности на микролитр, инкубируют в течение 30-90 мин, предпочтительно при температуре 37°С, где в паре прямого и обратного праймеров, один праймер содержит детектируемую метку, которую используют для идентификации маркеров ГМИ, причем в качестве отрицательного контроля используют дифференцирующий олигонуклеотид SEQ ID NO: 36, частично комплементарный олигонуклеотиду SEQ ID NO: 8, а в качестве положительного контроля используют дифференцирующий олигонуклеотид SEQ ID NO: 34, которому комплементарен специфический олигонуклеотид SEQ ID NO: 35.
2. Способ по п.1, отличающийся тем, что исследуемые ДНК выделяют, по крайней мере, из одного образца, содержащего ГМИ, входящего в группу, состоящую из плодов или сока растений, зерна, муки или пищевых продуктов, в состав которых входят генетически модифицированные растения.
3. Способ по п.1, отличающийся тем, что определяемую метку выбирают из группы, состоящей из каталитических, лигандных, флуоресцентных или радиоактивных молекул, причем каталитическую молекулу выбирают из группы, включающей гемип, цианкобаламин или флавин, лигапдную молекулу выбирают из группы, включающей биотин, диоксигенин или динитробепзол, а флуоресцентную молекулу выбирают из группы, включающей FAM, TAMRA, Cy3, Cy5, Cy7, R6G, R110, ROX или JOE.
4. Способ по п.1, отличающийся тем, что детектирование и/или идентификацию ГМИ осуществляют путем сравнительного анализа уровня сигналов исследуемого образца, положительного и отрицательного контролей.
5. Специфический олигонуклеотид в качестве праймера симметричной мультипраймерпой ПЦР для способа идентификации маркеров генетически модифицированных растений по пп.1-4, который характеризуется нуклеотидной последовательностью, выбранной из последовательностей, представленных в SEQ ID NO: 4, 7, 8, 11-14, 21, 22.
6. Дифференцирующий олигонуклеотид в качестве зонда для идентификации маркеров генетически модифицированных растений для использования в способе идентификации ГМИ по пп.1-4, который характеризуется нуклеотидной последовательностью, выбранной из последовательностей, представленных в SEQ ID NO: 24, 28, 29, 33.
7. Биочип для идентификации маркеров ГМИ, представляющий собой несущий элемент, на котором иммобилизованы дифференцирующие олигонуклеотиды в виде зондов, формирующих кластеры, характеризующийся тем, что на его поверхности иммобилизированы более 6 типов кластеров с дифференцирующими олигонуклеотидами, выбранными из SEQ ID NO: 23-33, при этом биочип содержит, по меньшей мере, один кластер, включающий олигонуклеотид положительного контроля, представленный в SEQ ID NO: 34, и, по меньшей мере, один кластер, включающий олигонуклеотид отрицательного контроля, представленный в SEQ ID NO: 36.
8. Биочип по п.7, отличающийся тем, что несущий элемент чипа выполнен из материалов, входящих в группу, состоящую из полимеров, стекла, металлов, керамики или их комбинаций.
9. Биочип по п.8, отличающийся тем, что полимеры выбирают из группы, состоящей из полиметилметакрилата, полибутилметакрилата, поливинилхлорида, поликарбоната, сополимеров метилметакрилата и/или сополимеров бутилметакрилата с другими мономерами, такими как стирол, акрилонитрил.
10. Биочип по п.7, отличающийся тем, что несущий элемент чипа выполнен в виде плоских платформ, пленок с толщиной от 0,05 до 5,0 мм.
11. Биочип по п.7, отличающийся тем, что дополнительно содержит идентификатор, выполненный в виде штрихкода или элемента с магнитной записью.
12. Биочип по п.11, отличающийся тем, что идентификатор биочипа включает код номера партии и/или даты изготовления и информацию о перечне размещенных дифференцирующих олигонуклеотидов.
13. Набор для идентификации маркеров ГМИ, включающий:
а) по меньшей мере, один биочип для проведения разовой диагностики, содержащий более 6 типов дифференцирующих олигонуклеотидов, специфичных к идентифицируемым маркерам трансгенных растений, с последовательностями, представленными в SEQ ID NO: 23-33, один или более дифференцирующих олигонуклеотидов в качестве положительного контроля, с последовательностью, представленной в SEQ ID NO: 34, и один или более дифференцирующих олигонуклеотидов в качестве отрицательного контроля, с последовательностью, представленной в SEQ ID NO: 36,
б) реагенты, которые выбирают из реагентов для подготовки образца, реагентов для проведения мультипраймерной симметричной ПЦР, реагентов для проведения гибридизации и их комбинации и фермента экзонуклеазы,
в) специфические олигонуклеотиды, используемые в качестве праймеров ПЦР для идентификации маркеров ГМИ, последовательности которых представлены в SEQ ID NO: 1-22, и специфический олигонуклеотид, представленный в SEQ ID NO: 35, который комплементарен дифференцирующему олигонуклеотиду положительного контроля SEQ ID NO: 34, размещенному на биочипе в кластерах положительного контроля.
14. Комбинация олигонуклеотидов для выявления маркера NOS, преимущественно для идентификации 3' нетранслируемой области гена нопалинсинтазы, отличающаяся тем, включает олигонуклеотиды с последовательностями SEQ ID NO: 24 и SEQ ID NO: 3 и SEQ ID NO: 4.
15. Комбинация олигонуклеотидов для выявления маркера NPTII, преимущественно для идентификации гена, кодирующего неомицин-фосфотрансферазу II, отличающаяся тем, что включает олигонуклеотиды с последовательностями SEQ ID NO: 26 и SEQ ID NO: 7 и SEQ ID NO: 8.
16. Комбинация олигонуклеотидов для выявления маркера Lectin, преимущественно для идентификации гена семейства Le1 из Glycine max, отличающаяся тем, что включает олигонуклеотиды с последовательностями SEQ ID NO: 28 в качестве зонда и два специфических SEQ ID NO: 11 и SEQ ID NO: 12.
17. Комбинация олигонуклеотидов для выявления маркера Zein, преимущественно для идентификации гена семейства zein из Zea Mays, отличающаяся тем, что включает олигонуклеотиды с последовательностями SEQ ID NO: 29 и SEQ ID NO: 13 и SEQ ID NO: 14.
18. Комбинация олигонуклеотидов для выявления маркера Patatin, преимущественно для идентификации гена pat1 из Solanum tuberosum, кодирующего предшественник пататина, отличающаяся тем, что включает олигонуклеотиды с последовательностями SEQ ID NO: 33 и SEQ ID NO: 21 и SEQ ID NO: 22.
| НАБОР ПРАЙМЕРОВ ДЛЯ ДЕТЕКЦИИ И/ИЛИ ИДЕНТИФИКАЦИИ ТРАНСГЕННЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ ДНК В РАСТИТЕЛЬНОМ МАТЕРИАЛЕ И ЕГО СОДЕРЖАЩИХ ПРОДУКТАХ (ВАРИАНТЫ), ПРАЙМЕР (ВАРИАНТЫ), ПАРА ПРАЙМЕРОВ (ВАРИАНТЫ), СПОСОБ ДЕТЕКЦИИ И/ИЛИ ИДЕНТИФИКАЦИИ С ИХ ИСПОЛЬЗОВАНИЕМ (ВАРИАНТЫ) И УСТРОЙСТВО ДЛЯ ОСУЩЕСТВЛЕНИЯ СПОСОБА | 2004 |
|
RU2265668C1 |
| US 5518900, 21.05.1996 | |||
| Узел подвески газосборного колокола алюминиевого электролизера | 1991 |
|
SU1786195A1 |
| Установка для обеспыливания воздуха при классификации карбонатного песка | 1986 |
|
SU1420182A1 |
| US 6710171, 23.03.2004 | |||
| US 6492114, 10.12.2002 | |||
| US 6951722, 04.10.2005 | |||
| JP 2001333774 A, 04.12.2001 | |||
| СМАЗОЧНАЯ КОМПОЗИЦИЯ ДЛЯ ФОРМАТОРОВ- ВУЛКАНИЗАТОРОВ | 0 |
|
SU166799A1 |
| JP 2006345855 A, 28.12.2006 | |||
| US 6492114, 10.12.2002. | |||
Авторы
Даты
2012-06-20—Публикация
2007-09-14—Подача