Изобретение представляет собой биологический ДНК маркер для обнаружения геномной ДНК картофеля S. tuberosum и родственных дикорастущих видов рода Solanum sect. Petota в растительном материале и пищевых продуктах. Изобретение относится к молекулярной биологии и биотехнологии растений и представляет собой оригинальную нуклеотидную последовательность фрагмента гена Sus4, кодирующего сахарозосинтазу - ключевого фермента расщепления сахарозы в клубнях картофеля, и пары олигонуклеотидных ПЦР-праймеров для амплификации этого фрагмента ДНК.
Сахарозосинтаза (сахарозо-уридин дифосфат гликозилтрансфераза ЕС 2.4.1.13) - фермент, катализирующий обратимое превращение сахарозы в клубнях в присутствии УДФ в УДФ-глюкозу и фруктозу. Основная функция фермента заключается в обеспечении растения субстратом для синтеза крахмала. По данным секвенирования генома картофеля было показано, что фермент кодируется монокопийным геном Sus4, локализованным на 12 хромосоме картофеля, а его последовательность высоко специфична для генома картофеля (Potato Genome Sequencing Consortium, 2011).
Изобретение предназначено для качественного и количественного обнаружения геномных копий целевой (картофельной) ДНК (в том числе генетически модифицированной), как в растительном материале культивируемых сортов картофеля род Solanum, секция Petota, так и в продуктах его переработки (крупа, экстракты и проч.), путем проведения массового анализа методом полимеразной цепной реакции (ПЦР) и/или ПЦР с детекцией в режиме реального времени.
В настоящее время для определения присутствия ДНК заданного объекта, в том числе ГМО (генетически модифицированного объекта), в исследуемом материале, в том числе в пищевых продуктах и продуктах переработки растительного материала (крупа, экстракты и проч.), используют два альтернативных метода, основанных на качественной идентификации либо белков, либо нуклеиновых кислот.
Известен способ идентификации белков растений с использованием соответствующих антител (Stave J.W., Food Control, 1999, vol.10, pp.367-374). Известен метод обнаружения рекомбинантных чужеродных белков в продуктах питания с использованием специфических антител к данным белкам (патент №WO 0198523 (А2) - DETECTION METHOD OF GENETIC RECOMBINANT FOOD AND DETECTION KIT OF THAT, авторы PARK НЕЕ-YOUNG [KR], GD BIOTECH CO LTD [KR]; PARK НЕЕ YOUNG [KR], 2001).
Также известен метод определения присутствия ГМО на основе анализа карты расщепления протеазой рекомбинантных протеинов, которые экспрессируются в генетически модифицированных растениях (патент №KR 20020000127 (А) - DETECTION METHOD OF GENETICALLY MODIFIED ORGANISM AND DETECTION KIT THEREOF, автор PARK HUI YEONG [KR], GD BIOTECH CO LTD [KR], 2006).
Недостатками представленных выше методов является невозможность выявить рекомбинантные/чужеродные белки в пищевых продуктах, в растительном сырье и продуктах его переработки, прошедших термическую, химическую или микробиологическую обработку. Это связано с тем, что при химических и/или физических воздействиях белки объекта способны разлагаться и изменять свою конформацию, вторичную структуру. Кроме того, эти методы характеризуются низкой чувствительностью, занимают много времени и требуют значительных материальных затрат.
Наиболее близким к предлагаемому способу являются изобретения на основе детектирования нуклеиновых кислот (ДНК), специфичных для обнаружения геномных копий целевой ДНК объекта в тестируемом растительном материале, продуктах его переработки и пищевых продуктах. Данный подход предполагает проведение анализа с применением метода полимеразной цепной реакции (ПЦР)/ гибридизации со специфическими олигонулеотидными праймерами или ДНК-зондами, характерными для чужеродной ДНК. Специфичные олигонулеотидные праймеры или ДНК-зонды используются как для детекции непосредственно самой чужеродной (трансгенной) ДНК, так и в качестве эндогенного референсного контроля на наличие ДНК культурного растения - носителя ГМО в продуктах его переработки. Такого рода эндогенные референсные контроли представляют собой высокоспецифичные маркеры генов культурных растений.
В настоящее время известен метод количественного и качественного анализа генетически модифицированных растений (бобы, кукуруза) и продуктов их переработки на основе масс-спектрометрии и проведения rcPCR (real-competitive PCR) с использованием наборов специфических праймеров (патент №KR 20110126306, A METHOD FOR ANALYZING GMO USING COMPETITIVE PCR, авторы Lee Myung Hoon; Han Sang Mi; Lee Soo Bin; Lee Hyun Chul, D& P BIOTECH LTD.).
Также известен метод качественной и количественной детекции генетически модифицированного картофеля в продуктах его переработки с помощью праймеров и стандартной плазмиды. Для детекции используют наборы праймеров NLL, Cry3A, UGP-pb, в том числе праймеры на основе гена UDP-glucose phosphorylase (патент №KR 20040079053, PRIMER SETS AND PROBES FOR DETECTION OF GMO, AND STANDARD PLASMID FOR QUALITATIVE AND QUANTITATIVE DETECTION OF GMO TO EFFECTIVELY CARRY OUT QUALITATIVE AND QUANTITATIVE DETECTION OF GMO IN AGRICULTURAL PRODUCTS AND FOODS, авторы AHN IL PYEONG; KANG SANG HO; KIM TAE SAN; KIM YEONG MI; LEE THERESA; NOH JAE GYUN; PARK YONG HWAN, REPUBLIC KOREA MAN RURAL DEV., 2004).
Кроме того, в отечественной базе данных был выявлен ряд заявок на сходные изобретения.
Описывается способ идентификации последовательностей ДНК трансгенных растений в пищевых продуктах, предполагающий использование биочипа и специфичных олигонуклеотидов для выявления и амплификации маркера 35S промотора из вируса мозаики цветной капусты, маркера NOS - преимущественно для идентификации 3′ нетранслируемой области гена нопалинсинтазы, маркера OCS - преимущественно для идентификации 3′ нетранслируемой области гена октопинсинтазы, маркера NPTII - преимущественно для идентификации гена, кодирующего неомицин-фосфотрансферазу II, маркера GUS - преимущественно для идентификации гена, кодирущего бета-глюкуронидазу, маркера Lectin - преимущественно для идентификации гена семейства Lel из Glycine max, маркера Zein - преимущественно для идентификации гена семейства Zein из Zea mays, маркера bar - преимущественно для идентификации гена из Streptomyces hygroscopicus, кодирущего фосфотрицин-ацетилтрансферазу, маркера cry1Ab - преимущественно для идентификации гена из Bacillus thuringiensis, кодирующего Bt-токсин, маркера EPSPS - преимущественно для идентификации гена из Agrobacterium tumefaciens, кодирующего 5-енолпирувилшикимат-3-фосфатсинтазу и маркера Patatin - преимущественно для идентификации гена pat1, кодирующего предшественник пататина (заявка на патент 2007134194/13, ДИФФЕРЕНЦИРУЮЩИЙ И СПЕЦИФИЧЕСКИЙ ОЛИГОНУКЛЕОТИД ДЛЯ ИДЕНТИФИКАЦИИ ПОСЛЕДОВАТЕЛЬНОСТЕЙ ДНК ТРАНСГЕННЫХ РАСТЕНИЙ В ПИЩЕВЫХ ПРОДУКТАХ, СПОСОБ ИДЕНТИФИКАЦИИ ТРАНСГЕННЫХ ПРОДУКТОВ, БИОЧИП, КОМБИНАЦИЯ ОЛИГОНУКЛЕОТИДОВ (ВАРИАНТЫ) И НАБОР ДЛЯ ОСУЩЕСТВЛЕНИЯ ЭТОГО СПОСОБА, авторы Грановский И.Э., Белецкий И.П., Шляпникова Е.А., Шляпников Ю.М., Гаврюшкин А.В., Бирюков С.В., 14.09.2007).
Известен другой способ идентификации трансгенных последовательностей ДНК в растительном материале и продуктах на его основе с использованием специфических праймеров и флуоресцентно меченых зондов, комплементарных последовательностям чужеродной (трансгенной) ДНК (заявка на патент 2008146091/13, СПОСОБ ИДЕНТИФИКАЦИИ ТРАНСГЕННЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ ДНК В РАСТИТЕЛЬНОМ МАТЕРИАЛЕ И ПРОДУКТАХ НА ЕГО ОСНОВЕ, авторы - Абрамов Д.Д., Кузнецов В.В., Митрохин И.А., Цыдендамбаев В.Д. 24.11.2008).
Известен способ обнаружения генетически модифицированных организмов растительного происхождения методом ПЦР с использованием праймеров и флуоресцентно меченых зондов, специфичных для конкретного уникального трансформационного события, а именно для конкретной генетической конструкции, имеющей строго определенную локализацию в геномах генетически модифицированных организмов, промотору 35S вируса мозаики цветной капусты (cauliflower mosaic virus), геномной ДНК этого вируса, терминатору NOS агробактерии (Agrobacterium tumefasciens) или геномной ДНК этой агробактерии (заявка 2009123913/10 на патент СПОСОБ ВЫЯВЛЕНИЯ ГЕННО-ИНЖЕНЕРНО-МОДИФИЦИРОВАННЫХ ОРГАНИЗМОВ И ИСТОЧНИКОВ РАСТИТЕЛЬНОГО ПРОИСХОЖДЕНИЯ, НЕ ЗАРЕГИСТРИРОВАННЫХ В ′′ГОСУДАРСТВЕННОМ РЕЕСТРЕ ПИЩЕВЫХ ПРОДУКТОВ, МАТЕРИАЛОВ И ИЗДЕЛИЙ, РАЗРЕШЕННЫХ ДЛЯ ИЗГОТОВЛЕНИЯ НА ТЕРРИТОРИИ РОССИЙСКОЙ ФЕДЕРАЦИИ ИЛИ ВВОЗА НА ТЕРРИТОРИЮ РОССИЙСКОЙ ФЕДЕРАЦИИ И ОБОРОТА′′, ГЕНОМ КОТОРЫХ СОДЕРЖИТ ПРОМОТОР 35S И/ИЛИ ТЕРМИНАТОР NOS (авторы Абрамов Д.Д., Кузнецов В.В., Колин В.В, Митрохин И.А., Цыдендамбаев В.Д., 24.06.2009).
Таким образом, в патентных базах были выявлены патенты и заявки как на белковые, так и ДНК-маркеры для идентификации ГМО в объектах растительного происхождения, мониторинга пищевых продуктов, животных кормов и других товаров массового потребления, получаемых из различных культурных растений.
Белковые маркеры, как правило, нестабильны и методы детекции с их использованием отличаются невысокой чувствительностью. В свою очередь, ДНК маркеры высокоспецифичны. Но известные в настоящее время ДНК-маркеры, как правило, относятся либо к другим культурам, и не распространяют свое действие на картофель, либо базируются на последовательностях чужеродных ДНК, входящих в конструкцию (последовательностей cry1Ab, EPSPS, 35S промотора и др.), в то время как никаких специфичных референсных маркеров на присутствие геномной ДНК Solanum tuberosum и родственных видов Solanum секции Petota в патентах, касающихся методов анализа ГМО, описано не было.
В этом отношении высокоспецифичные для картофеля и других Solanum последовательности гена Sus4 представляют определенный интерес. Монокопийное состояние гена, а также высокая специфичность делает ДНК маркеры на его основе наиболее эффективными в качестве эндогенного референсного контроля при идентификации геномной ДНК картофеля, как в растительном материале (в том числе и ГМО), так и в продуктах его переработки.
Кроме того, маркер и система на его основе просты в применении и не требуют больших временных, трудовых и денежных затрат.
Задачей предлагаемого изобретения является обнаружение геномных копий целевой (картофельной) ДНК представителей рода Solanum, секции Petota в растительном материале, в том числе и генетически модифицированном, а также в продуктах переработки картофеля с использованием эндогенного референсного контроля - ДНК маркера гена Sus4 сахарозсинтазы путем проведения анализа методом ПЦР и/или ПЦР с детекцией в режиме реального времени.
Таким образом, данное изобретение - биологический ДНК-маркер, должно быть представлено:
а) олигонуклеотидными праймерами следующего состава:
SusF3a 5-CAAAAACAAACTGAACGAACATGCATTC-3
Sus4R: 5-GCGTTGACGTTCACACGGATG-3
которые используются для амплификации гена Sus4 и его гомологов в растительном материале и в продуктах переработки картофеля;
б) TaqMan-зондом следующего состава:
SusZp2 5′-FAM-AGCGATTGTTCTGCCCCCT-BHQ 3′
который используется для количественного определения гена Sus4 и его гомологов в растительном материале и в продуктах переработки картофеля по технологии TaqMan;
с) оригинальной нуклеотидной последовательностью 233 п.н. фрагмента гена картофеля Sus4
CAAAAACAAACTGAACGAACATGCATTCGAAGAACTCCTGAAAT CCACTCAGGTAATTTTGCTTTGTCTATATGTGTCACCAAAAGATCATAT ATGTATCATTTTTGAGTTTATATATGAATGCTACTATGATATGTTATATA CTAGGAAGCGATTGTTCTGCCCCCTTGGGTTGCACTTGCTATTCGTTTG AGGCCTGGTGTCTGGGAATACATCCGTGTGAACGTCAACGC
представляющей эндогенный референсный контроль, однозначно подтверждающий наличие геномной ДНК картофеля в исследуемом материале;
д) маркер должен быть специфичен для Solanum tuberosum и для родственных дикорастущих видов клубнеобразующего картофеля рода Solanum секции Petota;
е) маркер должен быть применим для обнаружения ДНК как культурного картофеля Solanum tuberosum, так и родственных дикорастущих видов клубнеобразующего картофеля рода Solanum секции Petota.
Вышеуказанный результат достигается тем, что использование биологического ДНК-маркера приводит к специфической амплификации фрагмента гена сахарозсинтазы Sus4 у сортов картофеля Solanum tuberosum и родственных видов клубнеобразующего картофеля, таким образом, подтверждая наличие геномных копий целевой (картофельной) ДНК.
Изобретение может найти применение при проведении санитарно-эпидемиологических экспертиз, сертификационных испытаний, а также при проведении производственного контроля качества растительного сырья и продуктов его переработки, что позволит обеспечить непрерывный мониторинг качества растительного сырья, продуктов питания и животных кормов и других товаров массового потребления, производимых из картофеля, на протяжении всей производственной цепи.
Методика использования изобретения.
С целью обнаружения целевой (картофельной) ДНК в объекте исследования собирают живой растительный материал известных сортов картофеля (индивидуальные растения 10-15 сортов Solanum tuberosum). Наилучшим материалом для последующего выделения ДНК являются молодые ткани листьев. При выделении ДНК из клубневого материала клубни предварительно проращивают до появления из глазковых почек 1-2 листков.
Также проводится экстракция ДНК из тестируемых продуктов его переработки (крахмал, детское питание, чипсы и др).
1. Экстракция ДНК из тестируемого объекта.
Выделение тотальной ДНК из тканей растений проводили по следующей методике.
1. Лизис клеток.
К листовой высечке (~0.05 мг) в пробирке типа Eppendorf объемом 1,5 мл добавляли 400 мкл лизирующего буфера (200 мМ трис-HCl, рН=7,5; 250 мМ NaCl; 25 мМ EDTA; 0,5% SDS). Растительный материал в буфере при комнатной температуре гомогенизировали с помощью специальных пестиков. Смесь инкубировали в термостате или водяной бане в течение 20 минут при 65°C.
2. Депротеинизация ДНК.
В пробирку со смесью, содержащей лизированные клетки и ядра, добавляли 200 мкл предварительно охлажденного 5М ацетата калия, перемешивали и инкубировали на ледяной бане 20 мин. После инкубации смесь центрифугировали 15 мин при 16000 g. Полученный надосадочный супернатант (около 450 мкл) переносили в новую пробирку и подвергали двукратной очистке равным объемом смеси фенол (содержит 0.1% антиоксидантов 8-оксихинолина и 0.2% меркаптоэтанола)/хлороформ (смесь хлороформа и изоамилового спирта (24:1, об/об)). Супернатант и смесь фенол/хлороформ плавно перемешивали до образования эмульсии, а затем центрифугировали 5 минут при 16000 g. После чего верхнюю фазу с растворенной ДНК отбирали пипеткой в чистую пробирку. Фенол-хлороформную депротеинизацию повторяли 2-3 раза до полной очистки раствора ДНК от белков.
3. Осаждение ДНК.
Осаждение ДНК производили путем добавления к очищенному супернатанту 0.6 объема изопропанола; смесь перемешивали и центрифугировали в течение 15 мин при 16000 g. Спирт сливали и осадок трижды промывали 1 мл 70% этанолом, просушивали на воздухе и растворяли в 20-25 мкл бидистиллированной стерильной воды.
Выделение ДНК из продуктов переработки картофеля
Объект исследования
В качестве объектов исследования использовали продукты, в состав которых входили картофель или пищевые добавки на основе картофеля. Вся продукция была получена из различных московских супермаркетов.
Экстракция ДНК
ДНК выделяли согласно (Булыгина и др., 2009). Небольшое количество образца (75-100 мг) суспендировали в 200 мкл буфера I (50 мМ трис-HCl рН 8.0; 5 мМ ЭДТА; 50 мкг/мл панкреатической РНКазы) с помощью гомогенизатора (Vortex, Германия) в течение 3-5 минут (до получения гомогенной суспензии), после чего к полученной суспензии добавляли 300 мкл лизирующего буфера II (0.2 М NaOH, 1% раствор додецилсульфата натрия, рН 12.0) и тщательно перемешивали путем пипетирования. Затем добавляли 300 мкл нейтрализующего буфера III (2.5 М раствор ацетата калия, рН 4.5) и повторно суспендировали в течение 20 с. Смесь центрифугировали при 11000 g в течение 3 мин. Супернатант подвергали дополнительной очистке с помощью набора реактивов Wizard DNAPreps (Promega, США) согласно рекомендациям производителя.
Экстракция ДНК из образцов с повышенным содержанием жиров
Образцы с повышенным содержанием жиров (>20%) предварительно обрабатывали хлороформом. Для этого на первом этапе выделения навеску образца растворяли в 500 мкл буфера I, добавляли 1 мл хлороформа и инкубировали 1 ч при 60°C. Смесь осаждали центрифугированием в течение 10 мин при 11000 g, после чего супернатант подвергали очистке, как описано выше. Концентрация ДНК в получаемых препаратах составляла 50-150 мкг/мл. РНК в препаратах присутствовала в следовых количествах - менее 1%, согласно данным электрофоретического анализа.
2. Оценка качества и количества экстрагированной ДНК.
Оценку качества и количества экстрагированной ДНК производят путем измерения оптической плотности раствора ДНК на спектрофотометре (Bio Photometer, Eppendorf или SmartSpec 3000, Bio-Rad, США) или при помощи аналитического электрофореза.
3. Полимеразная цепная реакция (ПЦР).
Классическую ПЦР реакцию проводят для предварительной оценки наличия целевой ДНК в объекте по следующей методике:
1. Проведение ПЦР
Выделенную ДНК использовали в качестве матрицы в реакции амплификации.
Амплификацию ДНК проводили в реакционной смеси объемом 15 мкл, содержащей 10x буфер для соответствующей полимеразы, 0,16 мМ каждого dNTP, 1.2 мМ MgCl2, 0.6 мкМ биологического ДНК маркера, 0.3 ед. DreamTaq полимеразы (Fermentas, Литва) и 100 нг геномной ДНК в ДНК-амплификаторе «Applied Biosystems 2700» (США) в режиме: денатурация - 30 с - 94°C; отжиг - 45 с - 57°C; синтез ДНК - 3 мин 20 с - 72°C (35 циклов) с предварительной денатурацией - 5 мин (94°C) и заключительной элонгацией PCR-фрагментов 10 мин - 72°C.
2. Электрофоретическое разделение и визуализация ПЦР-продуктов.
Продукты амплификации анализировали путем проведения аналитического электрофореза в 0.8% агарозном геле толщиной 5-7 мм, содержащем бромистый этидий в 1x ТВЕ буфере. В качестве маркеров молекулярного веса использовали Gene Ruler™ DNA Ladder Mix (′′Fermentas′′, Литва). Вхождение фрагментов в гель осуществляли при 50V в течение 10-20 минут, а разделение фрагментов - в течение 1-2 часов при 75-90V.
Визуализацию осуществляли путем окрашивания гелей бромистым этидием согласно инструкции производителя, с последующим фотодокументированием.
Положительным результатом считается получение уникального фрагмента ДНК размером около 233 п.н. Размер амплифицируемых продуктов определяют с помощью маркера молекулярных масс GeneRuler™ DNA Ladder mix (′′Fermentas′′, Литва).
Для количественного определения целевой ДНК в образце использовали метод ПЦР в режиме реального времени, по технологии TaqMan с использованием представленных выше олигонуклеотидных праймеров и TaqMan-зонда:
SusZp2 5′-FAM-AGCGATTGTTCTGCCCCCT-BHQ 3′
Реакцию проводили на ДНК-амплификаторе CFX96™ Real-Time PCR System (Bio-Rad, США) в 25 мкл 1x TaqMan ПЦР буфера (Синтол, Россия), содержащего 7,5 пкМ каждого праймера, 5 пкМ зонда и 20 нг целевой ДНК при температурном режиме: активация полимеразы 5 мин при 95°C, последующие 40 циклов - 25 с при 95°C, 60 с при 60°C.
Детекцию для каждого образца осуществляли в трехкратной повторности. В качестве отрицательного контроля (NTC-реакционная смесь без ДНК-матрицы) использовали ddH2O (Синтол, Россия).
4. Соответствие полученных фрагментов искомым последовательности гена Sus4, проверяется по следующей методике:
1. Клонирование полученных фрагментов в вектор.
Полученные продукты ПЦР реакции клонировали с использованием системы QIAGEN cloning plus kit (QIAGEN, Нидерланды). Лигирование с вектором проводили в 10 мкл реакционной смеси, которая включала 1 мкл вектора (pDrive Cloning Vector (50 нг/мкл)), 3 мкл ПЦР продукта, 5 мкл Ligation Master Mix и очищенную воду.
Для трансформации использовали QIAGEN EZ Competent Cells (из соответствующего набора). К 1 мл раствора, содержащего компетентные клетки, добавляли 2 мкл лигазной смеси и инкубировали на льду в течение 5 минут. Затем смесь нагревали на водяной бане при 42°C в течение 30 с, с последующим охлаждением на льду в течение 2 минут. К охлажденным клеткам добавляли 250 мкл среды SOC (из соответствующего набора). Полученную смесь разносили по чашкам Петри со средой LBA, содержащей ампицилин (100 мг/мл), XGal (40 мг/мл) и IPTG (100 мМ) по 100 мкл на чашку.
Отбор колоний на наличие вставки проводили при помощи метода цветной селекции. Чашки Петри выдерживали при температуре 37°C в течение 12 часов, затем отбирали белые колонии. Белые колонии используют в качестве матрицы в реакции амплификации.
2. Амплификация клонированных генов сахарозсинтазы Sus4
Амплификацию ДНК проводили в реакционной смеси объемом 15 мкл, содержащей 10x буфер для соответствующей полимеразы, 0,16 мМ каждого dNTP, 1.2 мМ MgCl2, 0.3 мкМ биологического ДНК маркера, представленного последовательностями RXUF и RXUR, 0.3 ед. DreamTaq полимеразы (Fermentas, Литва) и клетки бактериальной колонии (~0.3 мкл) в ДНК-амплификаторе «Applied Biosystems 2700» (США) в режиме: денатурация - 30 с - 94°C; отжиг праймера - 45 с - 57°C; синтез ДНК - 3 мин 20 с - 72°C (35 циклов) с предварительной денатурацией - 5 мин (94°C) и заключительной элонгацией PCR-фрагментов 10 мин - 72°C.
Применение праймерной пары должно приводить к образованию фрагмента ДНК, длиной 233 п.н.
Продукты амплификации анализировали с помощью аналитического электрофореза в 0.8% агарозном геле толщиной 5-7 мм, содержащем бромистый этидий в 1x ТВЕ буфере. В качестве маркеров молекулярного веса использовали Gene Ruler™ DNA Ladder Mix (Fermentas, Литва). Вхождение фрагментов в гель осуществляли при 50V в течение 10-20 минут, а разделение фрагментов - в течение 1-2 часов при 75-90V.
Первичные последовательности ДНК полученных ПЦР продуктов определяли секвенированием по методу Сэнгера на автоматическом анализаторе ДНК ABI3730 (Applied Biosystems, США).
Установленные последовательности ДНК анализировали при помощи программы BLAST (Altschul et al., 1990) на соответствие известным последовательностям функциональных генов сахарозсинтазы Sus4 картофеля.
Пример выполнения предлагаемого способа.
Способ идентификации гена сахарозсинтазы Sus4 у образцов картофеля.
Из растительного материала 15 сортов Solanum tuberosum, а также образцов 2 видов клубнеобразующего картофеля S. megistacrolobum, S. acaule получают ДНК с использованием вышеописанного метода. В качестве отрицательного контроля могут быть взяты растения любого вида, в качестве дополнительного отрицательного контроля можно использовать растения близкородственных видов из семейства Solanaceae (S. lycopersicum (томат), S. melongena (баклажан)). Эти виды предположительно несут наиболее схожие гомологичные последовательности гена Sus4 ввиду наибольшего эволюционно-филогенетического родства к Solanum tuberosum. Таким образом, отсутствие амплификации фрагмента у томата и баклажана будет подтверждать высокую специфичность проводимой реакции амплификации. ДНК из продуктов переработки получают описанным выше методом.
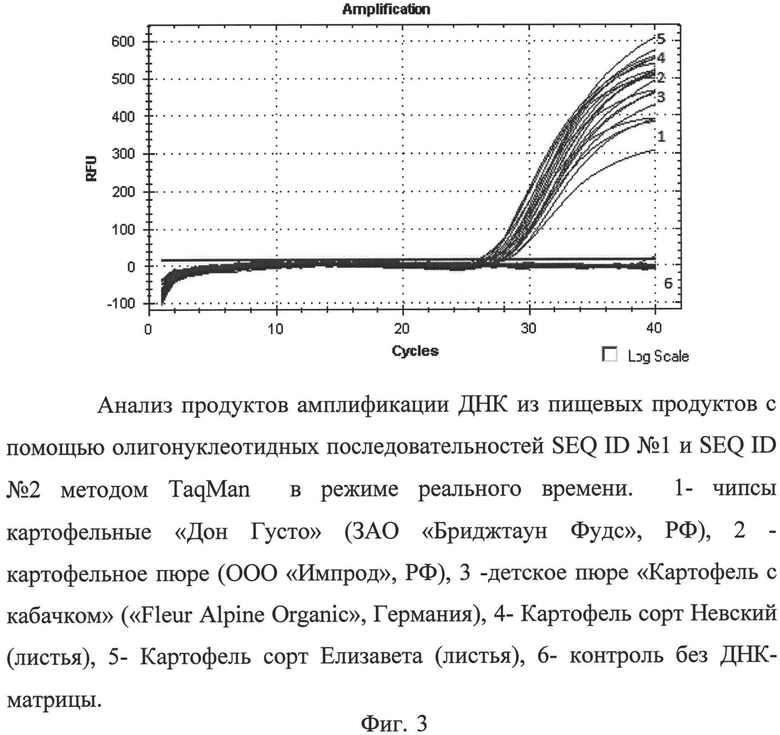
Далее ДНК, выделенную из каждого образца, используют в качестве матрицы для проведения полимеразной цепной реакции при описанных выше условиях с использованием биологического ДНК маркера, представленного последовательностями susF3a и susR4. Полученные продукты амплификации визуализируют путем электрофореза в геле. Размер полученных фрагментов у Solanum tuberosum и родственных дикорастущих видов клубнеобразующего картофеля рода Solanum секции Petota должен составлять ~233 п.н. (фиг. 1). Аналогичный фрагмент ДНК также будет амплифицироваться в материале, содержащем ДНК картофеля (фиг. 2, 3). Соответственно в любом другом материале, не содержащем ДНК картофеля, будет отсутствовать искомый фрагмент (фиг. 1).
Подтверждение природы амплифицированного фрагмента проводят путем его секвенирования на автоматическом анализаторе ДНК ABI3730 (Applied Biosystems, США). Соответствие полученных последовательностей гену Sus4 картофеля проверяется с использованием программы BLAST [Altschul et al, 1990] (фиг. 4).
Таким образом, использование биологического ДНК маркера приводит к специфической амплификации только фрагмента гена Sus4 картофеля, как из растений картофеля (в том числе и генетически модифицированных), так и из продуктов его переработки. Данный маркер может быть использован также в качестве эндогенного положительного контроля при проведении анализа на присутствие ГМО картофеля.
Литература.
1. Potato Genome Sequencing Consortium, Xu X, Pan S, Cheng S, Zhang B, Mu D, Ni P, Zhang G, Yang S, Li R, Wang J, Orjeda G, Guzman F, Torres M, Lozano R, Ponce O, Martinez D, De la Cruz G, Chakrabarti S K, Patil V U, Skryabin K G, Kuznetsov B B, Ravin N V, Kolganova T V, Beletsky A V, Mardanov A V, Di Genova A, Bolser D M, Martin D M, Li G, Yang Y, Kuang H, Hu Q, Xiong X, Bishop G J, Sagredo B, Mejía N, Zagorski W, Gromadka R, Gawor J, Szczesny P, Huang S, Zhang Z, Liang C, He J, Li Y, He Y, Xu J, Zhang Y, Xie B, Du Y, Qu D, Bonierbale M, Ghislain M, Herrera Mdel R, Giuliano G, Pietrella M, Perrotta G, Facella P, O′Brien K, Feingold S E, Barreiro L E, Massa G A, Diambra L, Whitty B R, Vaillancourt B, Lin H, Massa A N, Geoffroy M, Lundback S, DellaPenna D, Buell C R, Sharma S K, Marshall D F, Waugh R, Bryan G J, Destefanis M, Nagy I, Milbourne D, Thomson S J, Fiers M, Jacobs J M, Nielsen K L, Sønderkaer M, Iovene M, Torres G A, Jiang J, Veilleux R E, Bachem C W, de Boer J, Borm T, Kloosterman B, van Eck H, Datema E, Hekkert Bt, Goverse A, van Ham R C, Visser R G. Genome sequence and analysis of the tuber crop potato. Nature. 2011 Jul 10;475(7355):189-95. Altschul, S; Gish, W; Miller, W; Myers, E; Lipman, D (October 1990). ′′Basic local alignment search tool′′. Journal of Molecular Biology 215 (3):403-410.
2. Булыгина E.C., Колганова T.B., Сухачева M.B., Пантелеева А.Н., Патутина Е.О., Кузнецов Б.Б. Выделение ДНК из различных пищевых продуктов с помощью модифицированного щелочного метода. Биотехнология. №2, с.83-90, 2009.
| название | год | авторы | номер документа |
|---|---|---|---|
| БИОЛОГИЧЕСКИЙ ДНК МАРКЕР ДЛЯ ИДЕНТИФИКАЦИИ ГЕНА УСТОЙЧИВОСТИ К Х ВИРУСУ КАРТОФЕЛЯ | 2012 |
|
RU2522828C2 |
| ДНК-маркер для количественного определения геномной ДНК картофеля в растительном сырье и в продуктах, получаемых на его основе, в том числе при количественной идентификации ГМО | 2015 |
|
RU2658352C2 |
| БИОЛОГИЧЕСКИЙ ДНК МАРКЕР ДЛЯ ОПРЕДЕЛЕНИЯ СОРТОВ КАРТОФЕЛЯ, НАБОР И СПОСОБ СОРТОВОЙ ИДЕНТИФИКАЦИИ КАРТОФЕЛЯ | 2009 |
|
RU2413774C1 |
| ГЕН, ПРИДАЮЩИЙ УСТОЙЧИВОСТЬ К PHYTOPHTHORA INFESTANS (ФИТОФТОРОЗУ) В СЕМЕЙСТВЕ SOLANACEAE | 2003 |
|
RU2361920C2 |
| КЛОНИРОВАНИЕ И ИСПОЛЬЗОВАНИЕ ФУНКЦИОНАЛЬНОГО R-ГЕНА ИЗ SOLANUM CHACOENSE | 2010 |
|
RU2586509C2 |
| ДИФФЕРЕНЦИРУЮЩИЙ И СПЕЦИФИЧЕСКИЙ ОЛИГОНУКЛЕОТИДЫ ДЛЯ ИДЕНТИФИКАЦИИ ПОСЛЕДОВАТЕЛЬНОСТЕЙ ДНК ТРАНСГЕННЫХ РАСТЕНИЙ В ПИЩЕВЫХ ПРОДУКТАХ, СПОСОБ ИДЕНТИФИКАЦИИ ТРАНСГЕННЫХ ПРОДУКТОВ, БИОЧИП, КОМБИНАЦИЯ ОЛИГОНУКЛЕОТИДОВ (ВАРИАНТЫ) И НАБОР ДЛЯ ОСУЩЕСТВЛЕНИЯ ЭТОГО СПОСОБА | 2007 |
|
RU2453605C2 |
| ГЕНЫ И СПОСОБЫ ОБЕСПЕЧЕНИЯ УСТОЙЧИВОСТИ К ФИТОФТОРОЗУ ПАСЛЕНОВЫХ | 2008 |
|
RU2511423C2 |
| СПОСОБ ДИАГНОСТИКИ РАСТИТЕЛЬНОГО МАТЕРИАЛА НА ТРАНСГЕННОСТЬ | 2015 |
|
RU2607372C1 |
| СПОСОБЫ ОБНАРУЖЕНИЯ ДНК ДЛЯ САЙТ-СПЕЦИФИЧЕСКОЙ НУКЛЕАЗНОЙ АКТИВНОСТИ | 2013 |
|
RU2678001C2 |
| РЕКОМБИНАНТНАЯ ПОЛИНУКЛЕОТИДНАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ, ХАРАКТЕРИЗУЮЩАЯ УНИКАЛЬНЫЙ ТРАНСФОРМАЦИОННЫЙ АКТ МЕЖДУ ГЕНЕТИЧЕСКОЙ КОНСТРУКЦИЕЙ, ВКЛЮЧАЮЩЕЙ ГЕН cryIIIa, И ГЕНОМНОЙ ДНК КАРТОФЕЛЯ СОРТА ЕЛИЗАВЕТА, ЕЕ ПРИМЕНЕНИЕ И СОДЕРЖАЩИЕ ЭТУ ПОСЛЕДОВАТЕЛЬНОСТЬ КЛЕТКА, ТРАНСГЕННОЕ РАСТЕНИЕ И ЕГО ПОТОМСТВО | 2004 |
|
RU2286385C2 |

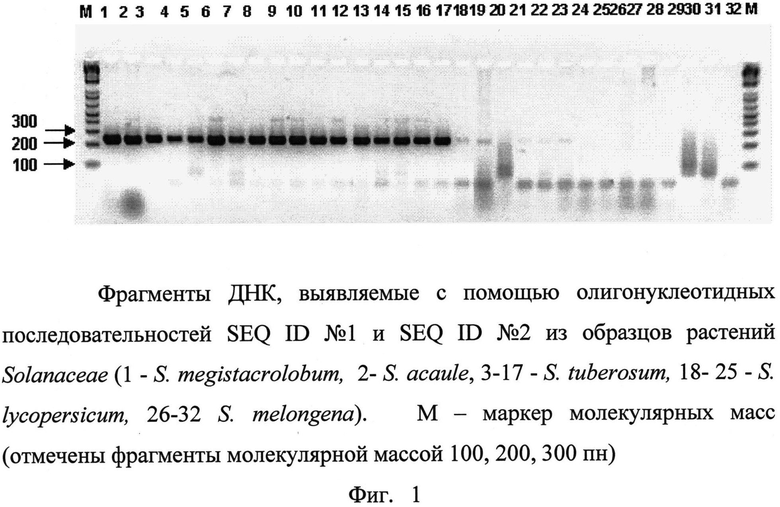


Изобретение относится к области биохимии, в частности к биологическому ДНК маркеру для обнаружения геномной ДНК картофеля S. tuberosum и родственных дикорастущих видов рода Solanum sect. Petota в растительном материале и пищевых продуктах, представляющий собой олигонуклеотидные праймеры - затравки полимеразной реакции SEQ ID №1 - 5′-CAAAAACAAACTGAACGAACATGCATTC-3′ SEQ ID №2 - 5′GCGTTGACGTTCACACGGATG-3′, позволяющие амплифицировать ген Sus4 у культурного картофеля S. tuberosum и родственных дикорастущих видов рода Solanum sect. Petota, а также в продуктах переработки картофеля, и таким образом идентифицировать ДНК картофеля. Изобретение позволяет эффективно обнаруживать геномную ДНК картофеля S. tuberosum и родственных дикорастущих видов рода Solanum sect. Petota в растительном материале и пищевых продуктах. 4 ил., 1 прим.
Биологический ДНК маркер для обнаружения геномной ДНК картофеля S. tuberosum и родственных дикорастущих видов рода Solanum sect. Petota в растительном материале и пищевых продуктах, представляющий собой олигонуклеотидные праймеры - затравки полимеразной реакции SEQ ID №1 - 5′-CAAAAACAAACTGAACGAACATGCATTC-3′ SEQ ID №2 - 5′GCGTTGACGTTCACACGGATG-3′, позволяющие амплифицировать ген Sus4 у культурного картофеля S. tuberosum и родственных дикорастущих видов рода Solanum sect. Petota, а также в продуктах переработки картофеля, и таким образом идентифицировать ДНК картофеля, при этом оригинальная нуклеотидная последовательность фрагмента гена картофеля Sus4 может быть использована в качестве референсного контроля для обнаружения целевой ДНК картофеля в тестируемых растениях и продуктах переработки картофеля и представляет собой: CAAAAACAAACTGAACGAACATGCATTCGAAGAACTCCTGAAA TCCACTCAGGTAATTTTGCTTTGTCTATATGTGTCACCAAAAGA TCATATATGTATCATTTTTGAGTTTATATATGAATGCTACTATG ATATGTTATATACTAGGAAGCGATTGTTCTGCCCCCTTGGGTTG CACTTGCTATTCGTTTGAGGCCTGGTGTCTGGGAATACATCCGTGTGAACGTCAACGC.
| БИОЛОГИЧЕСКИЙ ДНК МАРКЕР ДЛЯ ОПРЕДЕЛЕНИЯ СОРТОВ КАРТОФЕЛЯ, НАБОР И СПОСОБ СОРТОВОЙ ИДЕНТИФИКАЦИИ КАРТОФЕЛЯ | 2009 |
|
RU2413774C1 |
| Устройство для перекачивания серо углерода | 1930 |
|
SU21129A1 |
| UNTERGASSER A | |||
| et al., Primer3Plus, an enhanced web interface to Primer3, Nucleic Acids Research, 2007, Vol | |||
| Скоропечатный станок для печатания со стеклянных пластинок | 1922 |
|
SU35A1 |
| KR20110126306 A, 23.11.2011 | |||
| RU 2009123913 A, 27.12.2010 | |||
Авторы
Даты
2015-01-27—Публикация
2013-09-30—Подача