Область техники изобретения
Настоящее изобретение относится к локусам CRISPR Bifidobacterium animalis ssp. lactis и способам применения данных последовательностей.
Уровень техники изобретения
CRISPR (короткие палиндромные повторы, регулярно расположенные группами, Clustered Regularly Interspaced Short Palindromic Repeats) представляют собой характерный локус ДНК (то есть, набор или кластер последовательностей ДНК), обнаруженный в геномах многих бактерий и архей (для последнего обзора см., например, Sorek et al., «CRISPR - a widespread system that provides acquired resistance against phages in bacteria and archaea», Nature Reviews Microbiology, AOP, опубликованный в сети 24 декабря 2007 г.; doi:10.1038/nrmicro1793).
Недавно показано, что последовательности CRISPR могут функционировать в качестве разновидности «иммунной системы», которая помогает бактериям защищаться от фаговых инфекций (см., например, Barrangou et al., «CRISPR Provides Acquired Resistance Against Viruses in Prokaryotes», Science 315: 1709-12 (март 2007 г.); Deveau et al., J. Bacteriol. 190(4): 1390-1400 (февраль 2008 г.); Horvath et al., J. Bacteriol. 190(4): 1401-12 (февраль 2008 г.)). По меньшей мере восемь различных локусов CRISPR идентифицированы в геномах молочнокислых бактерий (см. Horvath et al., «Comparative analysis of CRISPR loci in lactic acid bacteria genomes», Int. J. Food Microbiol., электронная публикация 15 июля 2008 г.).
Кроме того, показано, что устойчивость к фагам у бактерий можно изменять путем введения последовательностей CRISPR в бактериальный геном. Например, удаление или добавление определенных последовательностей CRISPR от штаммов S. thermophilus приводит к появлению модифицированного устойчивого к фагу фенотипа (см., например, Barrangou et al. 2007 выше; Deveau et al., 2008, выше). В международной публикации № WO 2007/025097 A2, опубликованной 1 марта 2007 г. (которая включена в данный документ посредством ссылки), в числе прочего раскрыто использование локусов CRISPR для изменения устойчивости бактериального штамма к экзогенной нуклеиновой кислоте (например, фаговой инфекции).
Структура локуса CRISPR включает ряд коротких повторяющихся последовательностей, называемых «повторами». Повторы находятся в кластерах и в одном локусе CRISPR идентифицировано вплоть до 249 повторов (см., например, Sorek et al., 2007, выше), как правило, они правильно чередуются с уникальными вставочными последовательностями, называемыми «спейсерами». Обычно длина повторов CRISPR варьирует от примерно 24 до 47 п.о., и они являются частично палиндромными (см. Sorek et al., 2007, выше). Повторы, как правило, собраны в кластеры (вплоть до примерно 20 или более на геном) из повторяющихся единиц (см. Sorek et al., 2007, выше). Спейсеры расположены между двумя повторами и, как правило, каждый спейсер обладает уникальными последовательностями длиной примерно 20-72 п.о. (см. Sorek et al., 2007, выше). Многие спейсеры идентичны или имеют высокую степень гомологии с известными последовательностями фагов. Было показано, что вставка последовательности спейсера из конкретного фага в CRISPR бактерий может придавать устойчивость к данному фагу (см., например, Barrangou et al., «CRISPR Provides Acquired Resistance Against Viruses in Prokaryotes», Science 315: 1709-12 (март 2007 г.).
Помимо повторов и спейсеров, локус CRISPR также включает лидерную последовательность и, часто, набор из двух-шести связанных cas-генов. Лидерная последовательность, как правило, представляет собой AT-богатую последовательность длиной вплоть до 550 п.о., непосредственно примыкающую к 5'-концу первого повтора (см. Sorek et al., 2007, выше). Новая единица «повтор-спейсер» почти всегда добавляется к локусу CRISPR между лидерной последовательностью и первым повтором (см., например, Sorek et al., 2007, выше). Однако установлено, что приобретение устойчивости к фагу также может происходить в связи с добавлением нового спейсера и сопутствующим удалением спейсера из лидерной последовательности CRISP (см., например, Deveau et al., выше).
Считается, что белки, кодируемые связанными cas-генами, действуют как бактериальная «иммунная система», что придает устойчивость против фагов. Было высказано предположение, что набор из последовательностей «повтор-спейсер» транскрибируется в длинную РНК и повторы способствуют образованию вторичной структуры, которую cas-белки узнают и процессируют с образованием малых РНК, действующих по механизму, подобному РНК-интерференции (см. Sorek et al., 2007, выше). Brouns et al. (2008) сообщали, что комплекс из пяти cas-белков (CasA, CasB, CasC, CasD и CasE) в CRISPR/cas системе E. coli K12, названный «каскадом», расщепляет предшественник РНК CRISPR в каждом повторе и сохраняет продукт расщепления, содержащий происходящую из вируса последовательность. Предполагается, что с помощью Cas3-геликазы эти зрелые РНК CRISPR затем служат в качестве небольших направляющих РНК, которые позволяют каскаду препятствовать распространению вируса (см., например, Brouns et al., «Small CRISPR RNAs Guide Antiviral Defense in Prokaryotes», Science 321: 960-964 (2008)).
Последовательности CRISPR относятся к числу наиболее быстро развивающихся геномных структур у бактерий. Вследствие этого, а также из-за относительной простоты их последовательности (то есть, повтор-спейсер-повтор), последовательности CRISPR представляют собой идеальную геномную систему для обнаружения, типирования и отслеживания определенных штаммов бактерий. Способы использования последовательностей CRISPR для обнаружения, типирования и отслеживания штаммов бактерий раскрыты, например, в опубликованной патентной заявке США 2006/01990190 A1, опубликованной 7 сентября 2006 г., которая включена в данный документ посредством ссылки.
Локус CRISPR также представляет собой очень удобную, надежную, естественную и легко выявляемую геномную систему маркировки, которая не влияет на другие физиологические свойства маркированных прокариот. Способы использования известного фага для индукции CRISPR-метки (например, добавление единицы «повтор-спейсер») в бактериальном штамме раскрыты, например, в опубликованной патентной заявке США 2008/0124725 A1, опубликованной 29 мая 2008 г., которая включена в данный документ посредством ссылки.
Сущность изобретения
В некоторых вариантах осуществления изобретение относится к выделенной нуклеиновой кислоте, содержащей последовательность локуса CRISPR бифидобактерий, выбранную из группы, состоящей из BalalBala1 (SEQ ID NO: 1), CRISPRo9a (SEQ ID NO: 25), CRISPRo9b (SEQ ID NO: 26), CRISPRo91 (SEQ ID NO: 27), CRISPRo164 (SEQ ID NO: 28), CRISPRo228 (SEQ ID NO: 29), CRISPRo245 (SEQ ID NO: 30) и CRISPRo327 (SEQ ID NO: 31).
В некоторых вариантах осуществления изобретение относится к выделенной нуклеиновой кислоте, содержащей последовательность повтора BalalBala1, выбранную из SEQ ID NO: 2 и ее вариантных последовательностей. В некоторых вариантах осуществления варианты последовательности SEQ ID NO: 2 могут быть выбраны из замены Т на С в положении 12, замены Т на С в положении 14 и замены А на G в положении 36.
В некоторых вариантах осуществления изобретение относится к выделенной нуклеиновой кислоте, содержащей последовательность спейсера BalalBala1, выбранную из SEQ ID NO: 3-24. В некоторых вариантах осуществления последовательность спейсера BalalBala1 выбрана из SEQ ID NO: 13, 14 и 15.
В некоторых вариантах осуществления изобретение относится к выделенной нуклеиновой кислоте, содержащей последовательность
единицы «повтор-спейсер» Bala1, в которой последовательность повтора представляет собой SEQ ID NO: 2 и последовательность спейсера Bala1 выбрана из SEQ ID NO: 3-24.
В некоторых вариантах осуществления изобретение относится к выделенной нуклеиновой кислоте, содержащей последовательность повтора CRISPRo, выбранную из SEQ ID NO: 32-42, 53-60, 68-75, 83-92, 102-109, 117-125 и 134-143. В других вариантах осуществления изобретение относится к выделенной нуклеиновой кислоте, содержащей последовательность спейсера CRISPRo, выбранную из SEQ ID NO: 43-52, 61-67, 76-82, 93-101, 110-116, 126-133 и 144-152.
В некоторых вариантах осуществления изобретение относится к выделенной нуклеиновой кислоте, содержащей последовательность единицы «повтор-спейсер» CRISPRo, в которой последовательность повтора выбрана из SEQ ID NO: 32-42, 53-60, 68-75, 83-92, 102-109, 117-125 и 134-143, а последовательность спейсера выбрана из SEQ ID NO: 43-52, 61-67, 76-82, 93-101, 110-116, 126-133 и 144-152.
В других вариантах осуществления изобретение также относится к выделенным нуклеиновым кислотам, содержащим последовательность, способную гибридизоваться в строгих условиях с любой из описанных выше выделенных нуклеотидных последовательностей Bala1 или CRISPRo.
В некоторых вариантах осуществления изобретения любую из описанных выше выделенных нуклеотидных последовательностей Bala1 или CRISPRo можно встраивать в вектор. Таким образом, изобретение относится к вектору, содержащему локус CRISPR, выбранный из группы, состоящей из Bala1 (SEQ ID NO: 1), CRISPRo9a (SEQ ID NO: 25), CRISPRo9b (SEQ ID NO: 26), CRISPRo91 (SEQ ID NO: 27), CRISPRo164 (SEQ ID NO: 28), CRISPRo228 (SEQ ID NO: 29), CRISPRo245 (SEQ ID NO: 30) и CRISPRo327 (SEQ ID NO: 31).
В других вариантах осуществления изобретение относится к вектору, содержащему любую из следующих последовательностей: последовательность повтора Bala1, выбранную из SEQ ID NO: 2 и ее вариантных последовательностей; последовательность спейсера Bala1, выбранную из SEQ ID NO: 3-24; и последовательность единицы «повтор-спейсер» Bala1, в которой последовательность повтора выбирают из SEQ ID NO: 2 и ее вариантов, а последовательность спейсера выбирают из SEQ ID NO: 3-24.
В других вариантах осуществления изобретение относится к вектору, содержащему любую из следующих последовательностей CRISPRo: последовательность повтора CRISPRo, выбранную из SEQ ID NO: 32-42, 53-60, 68-75, 83-92, 102-109, 117-125 и 134-143; последовательность спейсера CRISPRo, выбранную из SEQ ID NO: 43-52, 61-67, 76-82, 93-101, 110-116, 126-133 и 144-152; и последовательность единицы «повтор-спейсер» CRISPRo, в которой последовательность повтора выбирают из SEQ ID NO: 32-42, 53-60, 68-75, 83-92, 102-109, 117-125 и 134-143, а последовательность спейсера выбирают из SEQ ID NO: 43-52, 61-67, 76-82, 93-101, 110-116, 126-133 и 144-152.
В некоторых вариантах осуществления изобретение относится к рекомбинантному штамму бактерий с измененной устойчивостью к фагу, содержащему любую из следующих последовательностей Bala1: последовательность повтора Bala1, выбранную из SEQ ID NO: 2 и ее вариантов; последовательность спейсера Bala1, выбранную из SEQ ID NO: 3-24; и последовательность единицы «повтор-спейсер» Bala1, в которой последовательность повтора выбирают из SEQ ID NO: 2 и ее вариантов, а последовательность спейсера выбирают из SEQ ID NO: 3-24.
В некоторых вариантах осуществления изобретение относится к рекомбинантному штамму бактерий с измененной устойчивостью к фагу, содержащему любую из следующих последовательностей CRISPRo: последовательность повтора CRISPRo, выбранную из SEQ ID NO: 32-42, 53-60, 68-75, 83-92, 102-109, 117-125 и 134-143; последовательность спейсера CRISPRo, выбранную из SEQ ID NO: 43-52, 61-67, 76-82, 93-101, 110-116, 126-133 и 144-152; и последовательность единицы «повтор-спейсер» CRISPRo, в которой последовательность повтора выбирают из SEQ ID NO: 32-42, 53-60, 68-75, 83-92, 102-109, 117-125 и 134-143, а последовательность спейсера выбирают из SEQ ID NO: 43-52, 61-67, 76-82, 93-101, 110-116, 126-133 и 144-152.
В некоторых вариантах осуществления вышеуказанных рекомбинантных штаммов бактерии представляют собой бифидобактерии, а в одном варианте осуществления - B. lactis.
В некоторых вариантах осуществления настоящее изобретение относится к способам получения рекомбинантных штаммов бактерий с измененной устойчивостью к фагу, включающим (a) трансформирование бактерий нуклеиновой кислотой, содержащей последовательность спейсера Bala1, выбранную из SEQ ID NO: 3-24; (b) контактирование трансформированных бактерий с фагом; и (c) выделение трансформированных бактерий, проявляющих устойчивость к фагу. В некоторых вариантах осуществления рекомбинантный штамм бактерий обладает повышенной устойчивостью к фагу (например, полной устойчивостью), а в других вариантах осуществления устойчивость к фагу понижена (например, устойчивость к фаговой инфекции отсутствует). В некоторых вариантах осуществления способа нуклеиновая кислота содержит локус Bala1 или CRISPRo, имеющий последовательность, выбранную из Bala1 (SEQ ID NO: 1), CRISPRo9a (SEQ ID NO: 25), CRISPRo9b (SEQ ID NO: 26), CRISPRo91 (SEQ ID NO: 27), CRISPRo164 (SEQ ID NO: 28), CRISPRO228 (SEQ ID NO: 29), CRISPRo245 (SEQ ID NO: 30) и CRISPRo327 (SEQ ID NO: 31). В других вариантах осуществления способа нуклеиновая кислота содержит последовательность, выбранную из последовательности повтора Bala1 (SEQ ID NO: 2 и ее варианты), последовательности спейсера Bala1 (SEQ ID NO: 3-24) и последовательности единицы «повтор-спейсер», в которой последовательность повтора выбирают из SEQ ID NO: 2 и ее вариантов, а последовательность спейсера выбирают из SEQ ID NO: 3-24. В других вариантах осуществления способа нуклеиновая кислота содержит последовательность, выбранную из последовательности повтора CRISPRo, выбранной из SEQ ID NO: 32-42, 53-60, 68-75, 83-92, 102-109, 117-125 и 134-143; последовательности спейсера CRISPRo, выбранной из SEQ ID NO: 43-52, 61-67, 76-82, 93-101, 110-116, 126-133 и 144-152; и последовательности единицы «повтор-спейсер» CRISPRo, в которой последовательность повтора выбирают из SEQ ID NO: 32-42, 53-60, 68-75, 83-92, 102-109, 117-125 и 134-143, а последовательность спейсера выбирают из SEQ ID NO: 43-52, 61-67, 76-82, 93-101, 110-116, 126-133 и 144-152.
В некоторых вариантах осуществления настоящее изобретение относится к способам типирования штаммов, обнаружения штаммов и/или отслеживания штаммов, включающим (a) амплификацию геномной ДНК из представляющего интерес штамма при помощи по меньшей мере одной пары праймеров, где указанная геномная ДНК содержит по меньшей мере часть последовательности локуса CRISPR; (b) обнаружение ампликона, полученного на этапе (a), где указанный обнаруженный ампликон служит признаком конкретного представляющего интерес штамма. В некоторых вариантах осуществления способа обнаружение ампликона производят методом, выбранным из измерения относительного размера с помощью гель-электрофореза (например, агарозных гелей) или масс-спектрометрического анализа; гибридизации с зондами известной последовательности (например, иммобилизованными зондами на микрочипе) и секвенирования (например, определения частичной или полной последовательности ампликона). В некоторых вариантах осуществления способа каждый праймер из пары комплементарен по меньшей мере части последовательности локуса CRISPR. В некоторых вариантах осуществления один праймер из пары комплементарен первому повтору и другой праймер из пары комплементарен концевому повтору локуса CRISPR, в результате чего полученный ампликон содержит весь локус CRISPR, или по меньшей мере часть каждого повтора и спейсера локуса CRISPR. В некоторых вариантах осуществления пара праймеров включает SEQ ID NO: 153 и 154.
В некоторых вариантах осуществления способов типирования штаммов, обнаружения штаммов и/или отслеживания штаммов каждый праймер из пары комплементарен части геномной ДНК таким образом, что пара праймеров амплифицирует по меньшей мере часть локуса CRISPR. Как правило, указанные части геномной ДНК будут содержать последовательности в непосредственной близости от и/или часть последовательности локуса CRISPR. В некоторых вариантах осуществления способов каждый праймер из пары комплементарен по меньшей мере части последовательности повтора локуса CRISPR, в результате чего при амплификации получают ампликон, содержащий по меньшей мере одну последовательность спейсера локуса CRISPR. В некоторых вариантах осуществления способов последовательности пары праймеров выбирают так, что каждый праймер комплементарен по меньшей мере части другого конца (то есть, либо 5', либо 3') последовательности повтора локуса CRISPR, в результате чего при амплификации получают множество ампликонов с последовательностями множества спейсеров, расположенных между повторами локуса CRISPR. В таком варианте осуществления множество ампликонов обнаруживают путем секвенирования или гибридизации со множеством зондов, комплементарных последовательностям спейсеров. В одном варианте осуществления способа множество ампликонов гибридизуют со множеством иммобилизованных зондов (например, микрочипом), в результате чего множество обнаруженных последовательностей спейсеров является признаком конкретного представляющего интерес штамма.
В некоторых вариантах осуществления способов типирования штаммов, обнаружения штаммов и/или отслеживания штаммов комплементарная последовательность локуса CRISPR представляет собой последовательность повтора, выбранную из SEQ ID NO: 2, 32-42, 53-60, 68-75, 83-92, 102-109, 117-125 и 134-143. В других вариантах осуществления способа каждый праймер из пары комплементарен по меньшей мере части последовательности спейсера локуса CRISPR, а в некоторых вариантах осуществления каждый праймер из пары комплементарен по меньшей мере части последовательности другого спейсера, в результате чего полученный ампликон содержит по меньшей мере одну последовательность повтора, расположенную между двумя последовательностями спейсеров. В некоторых вариантах осуществления способа один праймер из пары комплементарен последовательности спейсера, прилегающей к первому повтору локуса CRISPR. В некоторых вариантах осуществления один праймер из пары комплементарен последовательности спейсера, прилегающей к первому повтору локуса CRISPR, а другой праймер из пары комплементарен последовательности спейсера, прилегающей к концевому повтору локуса CRISPR, в результате чего полученный ампликон содержит все или по меньшей мере часть каждого спейсера в локусе CRISPR.
В некоторых вариантах осуществления способов типирования штаммов, обнаружения штаммов и/или отслеживания штаммов локус CRISPR выбирают из группы, состоящей из Bala1 (SEQ ID NO: 1), CRISPRo9a (SEQ ID NO: 25), CRISPRo9b (SEQ ID NO: 26), CRISPRo91 (SEQ ID NO: 27), CRISPRo164 (SEQ ID NO: 28), CRISPRo228 (SEQ ID NO: 29), CRISPRo245 (SEQ ID NO: 30) и CRISPRo327 (SEQ ID NO: 31).
В некоторых вариантах осуществления настоящее изобретение относится к набору для типирования штаммов, обнаружения штаммов и/или отслеживания штаммов, включающему упакованный комплект из (a) контейнера с композицией реагентов для амплификации, содержащей ДНК-полимеразу, буфер амплификации и по меньшей мере одну пару праймеров, где каждый праймер из пары комплементарен части геномной ДНК так, что пара праймеров способна амплифицировать по меньшей мере часть последовательности повтора или спейсера локуса CRISPR; и (b) контейнера с композицией реагентов для обнаружения, содержащей зонд, способный гибридизоваться в строгих условиях по меньшей мере с частью локуса CRISPR, амплифицированной с помощью пары праймеров. В некоторых вариантах осуществления каждая пара праймеров из набора комплементарна по меньшей мере части последовательности повтора или спейсера локуса CRISPR. В некоторых вариантах осуществления каждый праймер из пары в наборе комплементарен части геномной ДНК, так что пара праймеров способна амплифицировать по меньшей мере часть локуса CRISPR, выбранного из группы, состоящей из Bala1 (SEQ ID NO: 1), CRISPRo9a (SEQ ID NO: 25), CRISPRo9b (SEQ ID NO: 26), CRISPRo91 (SEQ ID NO: 27), CRISPRo164 (SEQ ID NO: 28), CRISPRo228 (SEQ ID NO: 29), CRISPRo245 (SEQ ID NO: 30) и CRISPRo327 (SEQ ID NO: 31). В некоторых вариантах осуществления пара праймеров включает SEQ ID NO: 153 и 154.
В некоторых вариантах осуществления изобретение относится к способу маркировки штамма бифидобактерий, включающему (a) подвергание родительского штамма бифидобактерий действию фага; (b) отбор нечувствительного к фагу мутанта и (c) сравнение последовательности локуса CRISPR или ее части из родительского штамма и нечувствительного к фагу мутантного штамма, в результате чего наличие дополнительной единицы «повтор-спейсер» в последовательности локуса CRISPR нечувствительного к фагу мутанта указывает на то, что штамм маркирован. В некоторых вариантах осуществления способа маркировки штамма локус CRISPR выбирают из группы, состоящей из Bala1 (SEQ ID NO: 1), CRISPRo9a (SEQ ID NO: 25), CRISPRo9b (SEQ ID NO: 26), CRISPRo91 (SEQ ID NO: 27), CRISPRo164 (SEQ ID NO: 28), CRISPRO228 (SEQ ID NO: 29), CRISPRo245 (SEQ ID NO: 30) и CRISPRo327 (SEQ ID NO: 31).
В некоторых вариантах осуществления изобретение относится к выделенной нуклеиновой кислоте, кодирующей cas-ген локуса CRISPR Bala1. В некоторых вариантах осуществления cas-ген локуса CRISPR Bala1 кодирует аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 155, 156, 157, 158, 159 и 160. В одном варианте осуществления выбранная аминокислотная последовательность представляет собой любую из SEQ ID NO: 156, 158 и 159.
В некоторых вариантах осуществления изобретение относится к выделенной нуклеиновой кислоте, содержащей нуклеотидную последовательность, включающую два или более cas-генов, в которой cas-гены кодируют две или более аминокислотных последовательностей, выбранных из группы, состоящей из SEQ ID NO: 155, 156, 157, 158, 159 и 160. В одном варианте осуществления выделенная нуклеиновая кислота содержит все шесть cas-генов локуса CRISPR Bala1.
Краткое описание фигур
На фигуре 1 представлена нуклеотидная последовательность локуса Bala1 из генома B. lactis Bl-04 (SEQ ID NO: 1). 23 повтора, каждый из 36 п.о., которые начинаются с положения 1290, изображены полужирными буквами. 22 спейсера соответствуют записанным строчными буквами последовательностям, расположенным между каждой парой повторов. Подчеркнутые части соответствуют сайтам ПЦР-праймеров праймера 1 и праймера 2, использованных для получения 874 п.о. ампликона, как описано в примере 1.
На фигуре 2 представлены 23 повтора (R1-R23) в последовательности SEQ ID NO: 1 локуса Bala1, выровненные с каждым из 22 спейсеров (S1-S22).
На фигуре 3 представлено схематичное сравнение локуса CRISPR Bala1 из генома B. lactis Bl-04 и четырех других штаммов B. lactis (Bi-07, DSM10140, B420 и HN019). Локус CRISPR Bala1, как он появляется в геноме B. lactis с шестью расположенными ниже cas-генами, приведен сверху. Области «повтор-спейсер» из Bl-04 и DSM10140 показаны в развернутом виде чуть ниже с повторами, представленными в виде черных «бриллиантов», и спейсерами в виде пронумерованных прямоугольников. Лидерная последовательность локуса CRISPR представлена в виде белого прямоугольника, отмеченного буквой «L», и концевой повтор представлен в виде черного «бриллианта» с пометкой «T». Изображены все 22 единицы «повтор-спейсер» локуса CRISPR Bala1 для пяти штаммов. Три единицы «повтор-спейсер» (включающие спейсеры S10, S11 и S12), которые отсутствуют в штаммах DSM10140, B420 и HN019, четко обозначены как делеция.
На фигуре 4 представлено схематичное сравнение локуса CRISPR Bala1 из B. lactis Bl-04 с локусами CRISPR, обнаруженными в геномах семи других бифидобактерий, а также геномные схемы (B. longum NCC2705, B. longum DJO10A, B. dentium ATCC 27678 Bden1, B. dentium ATCC 27678 Bden2, B. catenlatum JCM1194 Bcat1, B. adolescentis ATCC 15703 Bado1a, B. adolescentis L2-32 Bado1b). Области «повтор-спейсер» изображены в виде черных прямоугольников, cas-гены представлены узкими стрелками, тогда как другие гены представлены прямоугольными стрелками. Прямоугольники вокруг стрелок указывают на то, что эти гены делетированы в другом штамме того же вида.
На фигуре 5 представлены аминокислотные последовательности шести Cas-белков, расположенные ниже области «повтор-спейсер» CRISPR Bala1, как схематично показано на фигуре 3. (A) последовательность фермента Cas1; (B) последовательность фермента Cas2; (C) последовательность белка Csb1, кодирующая предполагаемый CRISPR-ассоциированный csb-ген, расположенный непосредственно ниже cas2; (D) последовательность белка Csb2, кодирующая предполагаемый CRISPR-ассоциированный csb-ген, расположенный непосредственно ниже csb1, (E) последовательность фермента Cas3 и (F) последовательность белка Csb3, кодирующая предполагаемый CRISPR-ассоциированный csb-ген, расположенный непосредственно ниже cas3.
На фигуре 6 представлена последовательность локуса CRISPRo9a. Расположение рядов нуклеотидов скорректировано таким образом, что все одиннадцать повторов из 25 п.о. (полужирные заглавные буквы) выровнены, и последовательные номера добавлены для обозначения каждого из десяти спейсеров, исходя из очередности его появления в последовательности.
На фигуре 7 представлена последовательность локуса CRISPRo9b. Расположение рядов нуклеотидов скорректировано таким образом, что все восемь повторов из 25 п.о. (полужирные заглавные буквы) выровнены, и каждый из семи спейсеров обозначен последовательным номером, исходя из очередности его появления в последовательности.
На фигуре 8 представлена последовательность локуса CRISPRo91. Расположение рядов нуклеотидов скорректировано таким образом, что все восемь повторов из 25 п.о. (полужирные заглавные буквы) выровнены, и каждый из семи спейсеров обозначен последовательным номером, исходя из очередности его появления в последовательности.
На фигуре 9 представлена последовательность локуса CRISPRo164. Расположение рядов нуклеотидов скорректировано таким образом, что все десять повторов из 25 п.о. (полужирные заглавные буквы) выровнены, и каждый из девяти спейсеров обозначен последовательным номером, исходя из очередности его появления в последовательности.
На фигуре 10 представлена последовательность локуса CRISPRo228. Расположение рядов нуклеотидов скорректировано таким образом, что все восемь повторов из 25 п.о. (полужирные заглавные буквы) выровнены, и каждый из семи спейсеров обозначен последовательным номером, исходя из очередности его появления в последовательности.
На фигуре 11 представлена последовательность локуса CRISPRo245. Расположение рядов нуклеотидов скорректировано таким образом, что все девять повторов из 25 п.о. (полужирные заглавные буквы) выровнены, и каждый из восьми спейсеров обозначен последовательным номером, исходя из очередности его появления в последовательности.
На фигуре 12 представлена последовательность локуса CRISPRo327. Расположение рядов нуклеотидов скорректировано таким образом, что все десять повторов из 25 п.о. (полужирные заглавные буквы) выровнены, и каждый из девяти спейсеров обозначен последовательным номером, исходя из очередности его появления в последовательности.
Подробное описание изобретения
I. Обзор
Настоящее изобретение относится к локусам CRISPR, обнаруженным у вида Bifidobacterium animalis подвида lactis (называемого в данном документе «B. lactis») и к использованию нуклеотидных последовательностей данных локусов в различных способах применения, включая генную инженерию устойчивости к фагам, типирование и отслеживание штаммов, а также маркировку штаммов.
По меньшей мере два различных семейства локусов CRISPR идентифицировано в геноме B. lactis Bl-04BL-04: «Bala1» и «CRISPRo». Локус Bala1 сопровождается cas-генами, имеет высоко консервативные повторы и присутствует только однократно в геноме Bl-04Bl-04. Напротив, локус CRISPRo не сопровождается cas-генами, имеет вырожденные повторы (то есть, последовательности повторов, обладающие некоторой изменчивостью) и присутствует по меньшей мере в семи различных участках генома Bl-04Bl-04.
Настоящее изобретение относится к композициям нуклеиновых кислот, способам и наборам, в которых используют нуклеотидные последовательности локусов Bala1 и CRISPRo, раскрытые в данном документе.
II. Определения
Все патенты и публикации, включая все последовательности, раскрытые в таких патентах и публикациях, на которые ссылаются в данном документе, специально включены посредством ссылки. Если в данном документе не указано иное, все технические и научные термины, используемые в данном документе, имеют тот же смысл, который в них обычно вкладывает рядовой специалист в той области, к которой относится изобретение (см., например, Singleton et al., DICTIONARY OF MICROBIOLOGY AND MOLECULAR BIOLOGY, 2D ED, John Wiley and Sons, New York [1994]; и Hale and Marham, THE HARPER COLLINS DICTIONARY OF BIOLOGY, Harper Perennial, NY [1991], оба источника предоставляют специалисту основной словарь многих терминов, используемых в данном документе). Любые способы и материалы, аналогичные или эквивалентные различным вариантам осуществления, описанным в данном документе, можно использовать на практике или при тестировании настоящего изобретения.
Предполагается, что каждое максимальное (или минимальное) численное ограничение, раскрытое в данной спецификации, включает все меньшие (или большие) численные ограничения, как если бы такие меньшие (или большие) численные ограничения были специально прописаны в данном документе. Кроме того, подразумевается, что каждый числовой диапазон, раскрытый в данном описании, включает каждый более узкий числовой диапазон, находящийся в пределах более широкого числового диапазона, как если бы такие более узкие числовые диапазоны были специально прописаны в данном документе.
Используемые в данном документе формы единственного числа включают их множественное число, если из контекста ясно не следует иное. Так, например, ссылка на «клетку-хозяина» включает множество таких клеток-хозяев.
Предполагается, что используемое в данном документе словосочетание «по меньшей мере» при употреблении совместно со списком значений или терминов относится к каждому значению или термину в списке. Например, фразу «по меньшей мере 85%, 90%, 95% и 99% идентичности последовательности» используют для обозначения по меньшей мере 85%, по меньшей мере 90%, по меньшей мере 95% и/или по меньшей мере 99% идентичности последовательности.
Используемый в данном документе термин «содержащий» и родственные ему слова используют в их включающем смысле, то есть, эквивалентно термину «включающий» и соответствующим ему родственным словам.
Если не указано иное, нуклеиновые кислоты пишутся слева направо в ориентации от 5'- к 3'-концу; аминокислотные последовательности пишутся слева направо в ориентации от амино- к карбоксильному концу, соответственно. Разделы, представленные в данном документе, не являются ограничениями для различных аспектов или вариантов осуществления изобретения, которые можно воплощать, исходя из спецификации в целом. Соответственно, термины, определенные непосредственно ниже, более полно определены, исходя из спецификации в целом.
В данном документе при описании белков и кодирующих их генов обозначение гена, как правило, пишется курсивом. Обозначение белка, как правило, не пишется курсивом и, как правило, пишется с заглавной буквы.
Используемые в данном документе термины «выделенная» и «очищенная» означают молекулу (например, выделенную нуклеиновую кислоту) или другой компонент, который отделен от по меньшей мере одного другого компонента, с которым он связан естественным образом.
При использовании в данном документе выражение «производный из» охватывает выражения «происходящий из», «полученный из» или «выделенный из».
Используемый в данном документе термин «бактерии» означает любой из прокариотических микроорганизмов, существующих в виде отдельной клетки или в кластере или агрегате из отдельных клеток.
Используемый в данном документе термин «бифидобактерии» означает любой из видов грамположительных анаэробных разветвленных палочковидных бактерий, которые обычно составляют кишечную микрофлору и относятся к членам рода Bifidobacterium, включая, но не ограничиваясь ими, B. angulatum; B. animalis; B. asteroides; B. bifidum; B. boum; B. breve; B. catenulatum; B. choerinum; B. coryneforme; B. cuniculi; B. dentium; B. gallicum; B. gallinarum; B indicum; B. longum; B. magnum; B. merycicum; B. minimum; B. pseudocatenulatum; B. pseudolongum; B. psychraerophilum; B. pullorum; B. ruminantium; B. saeculare; B. scardovii; B. simiae; B. subtile; B. thermacidophilum; B. thermophilum; B. urinalis; B. sp.
При использовании в данном документе «B. lactis» означает Bifidobacterium animalis подвид lactis.
Используемый в данном документе термин «локус CRISPR» означает сегмент ДНК, включающий все из повторов и спейсеров CRISPR, начиная от первого нуклеотида первого повтора CRISPR и кончая последним нуклеотидом последнего (концевого) повтора CRISPR. Как правило, каждая последовательность спейсера в локусе CRISPR расположена между двумя повторами и, следовательно, локус содержит на одну последовательность больше повторов, чем спейсеров.
Используемые в данном документе термины «повтор CRISPR», «последовательность повтора» или «повтор» имеют обычный смысл, используемый в данной области - то есть, несколько коротких прямых повторяющихся последовательностей, демонстрирующих крайне незначительную или полное отсутствие вариаций последовательности в данном локусе CRISPR.
Используемые в данном документе термины «спейсер CRISPR», «последовательность спейсера» или «спейсер» означают неповторяющиеся последовательности, расположенные между повторами локуса CRISPR.
В некоторых вариантах осуществления настоящего изобретения «спейсер» означает сегмент нуклеиновой кислоты, фланкированный двумя повторами. Последовательности спейсеров CRISPR часто обладают значительной гомологией с природными последовательностями фагов или плазмид. Как правило, спейсеры расположены между двумя идентичными или почти идентичными последовательностями повторов. Таким образом, спейсеры часто идентифицируют путем анализа последовательностей сегментов ДНК, расположенных между двумя повторами CRISPR.
Используемый в данном документе термин «cas-ген» имеет свое обычное значение, используемое в данной области, где он означает один или более генов, которые присоединены, связаны, расположены рядом или в непосредственной близости от локуса CRISPR. Как правило, cas-гены, связанные с локусом CRISPR, кодируются нуклеотидами, расположенными в 5'-положении от лидерной последовательности CRISPR. Исчерпывающий обзор семейства cas-белков представлен Haft et al. (Haft et al., Comput. Biol., 1, 6 e60 [2005]; см. также Brouns et al. (2008) выше).
Используемые в данном документе термины «лидер CRISPR», «лидерная последовательность» или «лидер» означают некодирующую последовательность, расположенную непосредственно выше от 5'-конца локуса CRISPR. Как правило, лидерная последовательность CRISPR расположена между первым нуклеотидом первого повтора в локусе CRISPR и стоп-кодоном последнего cas-гена.
Используемый в данном документе термин «трейлер CRISPR» означает некодирующую последовательность, расположенную непосредственно ниже от 3'-конца локуса CRISPR - то есть, сразу после последнего нуклеотида последнего повтора CRISPR. Этот последний повтор CRISPR также называют «концевым повтором».
Используемый в данном документе термин «бактериофаг» или «фаг» имеет свое обычное значение, используемое в данной области, то есть, вирус, избирательно инфицирующий один или несколько видов бактерий.
Все используемые в данном документе термины «маркированные бактерии», «маркированная бактерия» и «меченые бактерии» используются взаимозаменяемо для обозначения бактерий, подвергнутых воздействию фага, и в которых один или несколько локусов CRISPR или их части были модифицированы таким образом, что бактерии стали устойчивыми к фагу. Как описано более подробно в данном документе, в некоторых вариантах осуществления маркированные бактерии подвергают воздействию более чем одного фага (например, либо итеративно, последовательно, либо одновременно), так что более одной геномной модификации накапливается в их локусах CRISPR таким образом, что они становятся нечувствительными к каждому из фагов, воздействию которых их подвергли.
Используемые в данном документе термины «измененный» или «изменения», используемые в контексте клеточной устойчивости к нуклеиновой кислоте, могут означать подавление, снижение, сокращение, индукцию, придание, восстановление, возрастание, увеличение или иное воздействие на устойчивость клетки к целевой нуклеиновой кислоте.
Используемый в данном документе термин «устойчивость к целевой нуклеиновой кислоте» означает, что устойчивость придана против любого элемента, который содержит или продуцирует целевую нуклеиновую кислоту или продукт ее транскрипции (например, клетки, фага, плазмид, «голой» ДНК). Типы элементов не ограничиваются живыми элементами, такими как клетки и фаги, но также включают неживые элементы, например, плазмиды или перемещающиеся элементы. Таким образом, в некоторых вариантах осуществления последовательности CRISPR по настоящему изобретению могут обеспечивать устойчивость против любого содержащего аминокислоту элемента и даже последовательностей свободной или «голой» нуклеиновой кислоты, которые включают целевую нуклеиновую кислоту. Устойчивость можно измерять с точки зрения выживания устойчивой клетки или с точки зрения предотвращения сохранения и/или выживания внесенной нуклеиновой кислоты (например, предотвращения репликации и/или транскрипции и/или экспрессии целевой нуклеиновой кислоты). Устойчивость не предназначена служить признаком того, что чужеродная ДНК обязательно встречает препятствие при проникновении в устойчивую клетку (то есть, при проникновении через клеточную мембрану). Более того, термин «устойчивость» не подразумевает, что клетка на 100% устойчива к целевой нуклеиновой кислоте или продукту ее транскрипции, но охватывает клетки, толерантные к целевой нуклеиновой кислоте или продукту ее транскрипции.
При использовании в данном документе «амплификация» означает производство дополнительных копий последовательности нуклеиновой кислоты. Амплификацию используют во многих способах применения последовательностей CRISPR (см., например, Mojica et al., «Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements», Journal of Molecular Evolution 60: 174-182 (2005); и Pourcel et al., «CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA and provide additional tools for evolutionary studies», Microbiology 151: 653-663 (2005)), включая варианты осуществления, раскрытые в данном документе (например, обнаружение, типирование, отслеживание и маркировка штаммов). В вариантах осуществления настоящего изобретения амплификацию, как правило, проводят хорошо известным специалистам в данной области методом «полимеразной цепной реакции» («ПЦР, PCR»). Кроме того, другие методы амплификации, включая, но не ограничиваясь ими, лигазную цепную реакцию («ЛЦР, LCR») и методы изотермальной амплификации, находят применение в настоящем изобретении. Хорошо известные методы изотермальной амплификации, применимые по настоящему изобретению, включают, но не ограничиваются ими, амплификацию замещением цепей (SDA), амплификацию Q-бета-репликазой, амплификацию, основанную на последовательности нуклеиновых кислот (NASBA), и самоподдерживающуюся репликацию последовательностей.
При использовании в данном документе «праймер» означает олигонуклеотид, существующий в природе (например, из очищенных фрагментов рестрикции) или полученный синтетическими методами, который способен действовать как точка инициации синтеза (например, для амплификации), будучи помещенным в условия, в которых индуцируется синтез продукта достройки праймера, комплементарного цепи нуклеиновой кислоты (например, ампликона) (то есть, при подходящей температуре, pH и в присутствии нуклеотидов и индуцирующего агента, такого как ДНК-полимераза). Праймеры для ПЦР могут состоять из рибонуклеотидов, дезоксирибонуклеотидов или их синтетических аналогов и, как правило, бывают длиной по меньшей мере примерно 10 нуклеотидов, и, наиболее типично, длиной по меньшей мере примерно 20 нуклеотидов. Методы составления режима и проведения ПЦР хорошо известны в данной области и включают, но не ограничиваются ими, методы с использованием парных праймеров, вложенных праймеров, одиночных специфических праймеров, вырожденных праймеров, ген-специфичных праймеров, вектор-специфичных праймеров, частично несовпадающих праймеров и так далее.
В различных вариантах осуществления настоящего изобретения пары праймеров используют в ПЦР для амплификации всего или части локуса CRISPR. В некоторых вариантах осуществления праймер может быть одноцепочечным, например, для достижения максимальной эффективности при амплификации, однако в других вариантах осуществления праймер может быть двухцепочечным. В некоторых вариантах осуществления праймер может представлять собой олигодезоксирибонуклеотид. Как правило, праймер должен быть олигонуклеотидом, достаточно длинным, чтобы служить затравкой для синтеза продуктов достройки в присутствии индуцирующего агента. Выбранная длина праймера, используемого в вариантах осуществления настоящего изобретения, будет зависеть от различных факторов, включая температуру, источник праймера и конкретное применение.
Используемый в данном документе термин «рекомбинантный» применительно к клетке, нуклеиновой кислоте или белку означает клетку, нуклеиновую кислоту или белок, модифицированные введением нуклеиновой кислоты или белка (либо родных, либо гетерологичных) при помощи вектора, либо происходящие из модифицированной таким образом клетки. Таким образом, «рекомбинантный штамм бактерий» означает бактериальный штамм, который модифицирован при помощи вектора для введения нуклеиновой кислоты (например, последовательности спейсера CRISPR) или белка.
Используемый в данном документе термин «вектор» означает любую молекулу нуклеиновой кислоты, в которую можно вставить другую молекулу нуклеиновой кислоты (например, последовательность единицы «повтор-спейсер» CRISPR) и которая может быть введена в клетки и реплицироваться в них. Таким образом, термин означает любую конструкцию нуклеиновой кислоты (и, при необходимости, любую связанную систему доставки), которую можно использовать для передачи генетического материала между различными клетками-хозяевами. Многие прокариотические векторы являются коммерчески доступными для производства рекомбинантных штаммов бактерий. Выбор соответствующих векторов находится в пределах компетенции специалистов в данной области.
Используемый в данном документе термин «плазмида» означает кольцевую двухцепочечную (ds) конструкцию ДНК, которую можно использовать в качестве вектора для введения ДНК в клетку. Плазмиды выступают в качестве экстрахромосомных самовоспроизводящихся генетических элементов во многих бактериях и у некоторых эукариот. В некоторых вариантах осуществления одну или несколько плазмид можно интегрировать в геном клетки-хозяина, в которую их ввели.
При использовании в данном документе «хозяин», «клетка-хозяин» или «штамм-хозяин» означают клетку, способную реплицировать и/или экспрессировать последовательность ДНК, введенную в клетку. В некоторых вариантах осуществления настоящего изобретения клетки-хозяева представляют собой бифидобактерии.
Используемый в данном документе термин «соответствующий родительский штамм» означает штамм, из которого происходит рекомбинантный штамм (например, исходный и/или штамм дикого типа). В некоторых вариантах осуществления соответствующий родительский штамм может быть штаммом, который сам был генетически изменен или модифицирован.
При использовании в данном документе «нуклеиновая кислота» означает нуклеотидную или полинуклеотидную последовательность и ее фрагменты или части, а также ДНК и РНК геномного или синтетического происхождения, которая может быть двухцепочечной или одноцепочечной, представляя собой либо смысловую, либо антисмысловую цепь.
При использовании в данном документе «гомологичная последовательность» означает нуклеотидную или полипептидную последовательность, имеющую по меньшей мере примерно 60%, по меньшей мере примерно 70%, по меньшей мере примерно 80%, по меньшей мере примерно 85%, по меньшей мере примерно 88%, по меньшей мере примерно 90%, по меньшей мере примерно 92%, по меньшей мере примерно 94%, по меньшей мере примерно 95%, по меньшей мере примерно 96%, по меньшей мере примерно 97%, по меньшей мере примерно 98%, по меньшей мере примерно 99% или даже большую идентичность последовательности с исследуемой нуклеотидной или аминокислотной последовательностью при оптимальном выравнивании для сравнения. В некоторых вариантах осуществления гомологичные последовательности имеют от примерно 80% до 100% идентичности последовательности, в некоторых вариантах осуществления - от примерно 90% до 100% идентичности последовательности и в некоторых вариантах осуществления - от примерно 95% до 100% идентичности последовательности.
Гомологию последовательностей можно определять стандартными методами, известными в данной области (см., например, Smith and Waterman, Adv. Appl. Math., 2: 482 [1981]; Needleman and Wunsch, J. Mol. Biol., 48: 443 [1970]; Pearson and Lipman, Proc. Natl. Acad. Sci. USA 85: 2444 [1988]; такие программы как GAP, BESTFIT, FASTA и TFASTA в пакете программ Wisconsin Genetics Software Package (Genetics Computer Group, Madison, WI); и Devereux et al., Nucl. Acid Res., 12: 387-395 [1984]). Полезные алгоритмы для определения гомологии последовательностей включают PILEUP и BLAST (Altschul et al., J. Mol. Biol., 215: 403-410 [1990]; и Karlin et al., Proc. Natl. Acad. Sci. USA 90: 5873-5787 [1993]). В PILEUP используется упрощение метода прогрессивного выравнивания Feng и Doolittle (Feng and Doolittle, J. Mol. Evol., 35: 351-360 [1987]). Метод аналогичен тому, который описан Higgins и Sharp (Higgins and Sharp, CABIOS 5: 151-153 [1989]). Полезные параметры PILEUP, включая стандартный штраф за открытие делеции, равный 3,00, стандартный штраф за удлинение делеции, равный 0,10, и штрафы за концевые делеции.
Особенно полезной программой BLAST является программа WU-BLAST-2 (см., Altschul et al., Meth. Enzymol, 266: 460-480 [1996]). В WU-BLAST-2 используются несколько параметров поиска, большинство из которых установлены на значения по умолчанию. Регулируемые параметры установлены со следующими значениями: протяженность совпадения = 1, разрыв совпадения = 0,125, порог слова (T) = 11. Параметры HSP S и HSP S2 являются динамическими значениями и устанавливаются самой программой в зависимости от состава конкретной последовательности и состава конкретной базы данных, против которой проводят поиск для интересующей последовательности. Однако значения могут быть скорректированы для повышения чувствительности. Значение % идентичности аминокислотной последовательности определяют путем деления числа совпадающих идентичных остатков на общее число остатков «более длинной» последовательности в выравниваемой области. Более длинной последовательностью является последовательность, имеющая наиболее актуальные остатки в выравниваемой области (делеции, внесенные программой WU-Blast-2 для максимального увеличения баллов выравнивания, игнорируются).
Используемый в данном документе термин «гибридизация» означает процесс, посредством которого цепь нуклеиновой кислоты связывается с другой цепью через комплементарное (например, по принципу Уотсона и Крика) спаривание оснований между цепями.
При использовании в данном документе «избирательно гибридизуемые» означает две нуклеиновые кислоты, имеющие последовательности, которые специфически гибридизуются друг с другом при умеренных и строгих условиях гибридизации и промывки. Условия гибридизации основаны на температуре плавления (Tm) связывающего нуклеиновую кислоту комплекса или зонда. Например, «максимальная строгость» условий, как правило, имеет место примерно при Tm-5°C (на 5° ниже, чем Tm зонда); «высокая строгость» - примерно при 5-10° ниже Tm; «промежуточная строгость» - примерно при 10-20°C ниже Tm зонда и «низкая строгость» - примерно при 20-25°C ниже Tm. Функционально, максимально строгие условия можно использовать для идентификации последовательностей, имеющих строгую идентичность или почти строгую идентичность с гибридизующимся зондом; тогда как гибридизацию с промежуточной или низкой строгостью можно использовать для идентификации или обнаружения гомологов полинуклеотидной последовательности.
Умеренные или высоко строгие условия гибридизации хорошо известны в данной области. Пример высоко строгих условий включает гибридизацию при примерно 42°C в 50% формамиде, 5X SSC, 5X растворе Денхардта, 0,5% SDS и 100 мкг/мл денатурированной ДНК-носителе, с последующим промыванием два раза в 2X SSC и 0,5% SDS при комнатной температуре и два дополнительных раза в 0,1X SSC и 0,5% SDS при 42°C. Пример умеренно строгих условий включает инкубацию в течение ночи при 37°C в растворе, содержащем 20% формамид, 5× SSC (150 мМ NaCl, 15 мМ цитрат тринатрия), 50 мМ фосфат натрия (pH 7,6), 5× раствор Денхардта, 10% декстрансульфат и 20 мг/мл денатурированной фрагментированной ДНК из спермы лосося, с последующим промыванием фильтров в 1× SSC примерно при 37-50°C. Специалистам в данной области известно, как должным образом регулировать температуру, ионную силу и так далее, чтобы они соответствовали таким факторам, как длина зонда и тому подобное.
Используемый в данном документе термин «введение» (и в прошедшем времени, «введенный»), употребляемый в контексте «введение последовательности нуклеиновой кислоты в клетку», относится к любому способу, подходящему для переноса последовательности нуклеиновой кислоты в клетку, включая, но не ограничиваясь ими, трансформацию, электропорацию, микроинъекцию в ядра, трансдукцию, трансфекцию (например, трансфекцию, опосредованную липофекцией и опосредованную DEAE-декстрином), инкубацию с кальций-фосфатным преципитатом ДНК, высокоскоростную баллистическую трансфекцию покрытыми ДНК микрочастицами, трансформацию, опосредованную агробактериями, и слияние протопластов.
В различных вариантах осуществления настоящего изобретения экзогенную нуклеиновую кислоту (например, конструкцию или вектор), содержащую последовательность локуса CRISPR или по меньшей мере ее часть, вводят в клетки бактериального штамма. Методы трансформирования бактериальных клеток нуклеиновыми кислотами, приводящие к интеграции в геном посредством, например, гомологичной рекомбинации, хорошо известны в данной области, например, см. Sambrook et al. (Molecular Cloning: A Laboratory Manual, 2е издание, 1989, Cold Spring Harbor Laboratory Press) и Ausubel et al., Current Protocols in Molecular Biology (1995), John Wiley & Sons, Inc. Трансформированные клетки можно культивировать в хорошо известных условиях для репликации и/или экспрессии интегрированной последовательности нуклеиновой кислоты в бактерии.
При использовании в данном документе «вносимая последовательность» означает последовательность ДНК, которую ввели в клетку-хозяина. Вносимая последовательность может представлять собой конструкцию ДНК, может представлять собой последовательность CRISPR, может кодировать один или несколько представляющих интерес белков (например, рекомбинантный вариант природного белка) и может включать фланкирующие последовательности, такие как промотор и терминатор, вокруг представляющего интерес белка. Например, вносимая последовательность может включать единицу «повтор-спейсер» Bala1.
При использовании в данном документе «фланкирующая последовательность» или «фланкирующая область» означает любую последовательность, находящуюся либо выше, либо ниже от обсуждаемой последовательности (например, каждый спейсер имеет повторы в качестве фланкирующих последовательностей). В некоторых вариантах осуществления фланкирующая последовательность присутствует только на одной стороне (либо на 3', либо на 5'), а в других вариантах осуществления она находится с каждой стороны фланкируемой последовательности.
Используемый в данном документе термин «интегрированная», употребляемый применительно к последовательности нуклеиновой кислоты, означает, что она встроена в хромосомную ДНК клетки-хозяина. В одном варианте осуществления настоящего изобретения рекомбинантную последовательность спейсера CRISPR, присущую бактериальным видам, встраивают в плазмиду, используемую для трансформации бактериальной клетки, которая не обладает этим спейсером в своем эндогенном геноме локусов CRISPR, и спейсер интегрируется в геномную ДНК трансформированной клетки.
III. Общие методы и варианты осуществления изобретения
В некоторых аспектах настоящее изобретение основывается на рутинных приемах и методах, применяемых в области генетической инженерии и молекулярной биологии. Следующие источники включают описание общей методологии, применяемой согласно данному изобретению: Sambrook et al., MOLECULAR CLONING: A LABORATORY MANUAL (2е издание, 1989); Kreigler, GENE TRANSFER AND EXPRESSION; A LABORATORY MANUAL (1990) и Ausubel et al., Eds. CURRENT PROTOCOLS IN MOLECULAR BIOLOGY (1994). В этих основных ссылках содержатся определения и методы, известные специалистам в данной области. Однако это не предполагает, что настоящее изобретение будет ограничено любыми конкретными описанными методами, протоколами и реагентами, поскольку они могут варьироваться.
A. Локус CRISPR из B. lactis
1. Идентификация и анализ локусов CRISPR в последовательности генома B. lactis Bl-04
Различные способы идентификации локусов CRISPR известны в данной области. Например, Jansen et al. описывают компьютерный подход, в котором в нуклеотидной последовательности ищут мотивы CRISPR при помощи программы PATSCAN на сервере отдела математики и компьютерных наук в Аргоннской национальной лаборатории, Argonne, III., USA (см., например, Jansen et al., «Identification of a novel family of sequence repeats among prokaryotes», OMICS 6: 23-33 (2002); Jansen et al., «Identification of genes that are associated with DNA repeats in prokaryotes», Molecular Microbiology 43: 1565-1575 (2002)). Примерный алгоритм, который можно использовать для идентификации мотивов CRISPR, был следующим p1=a … bc … dp1c … dp1c … dp1, где a и b представляли собой нижний и верхний предел размера последовательностей повторов, а p1 и c и d представляли собой нижний и верхний предел размера последовательностей спейсеров. Значения a, b, c и d могут варьироваться от примерно 15 до примерно 70 п.о. с шагом примерно 5 п.о. В некоторых вариантах осуществления локусы CRISPR идентифицируют при помощи точечных графиков (dotplots) (например, с помощью компьютерной программы Dotter).
Геном B. lactis Bl-04 был секвенирован при помощи комбинации из секвенирования по Сенгеру и пиросеквенирования. Полученные три контига были аннотированы с помощью набора программного обеспечения для биоинформатики ERGO™ (Integrated Genomics, Chicago, IL). Полная схематичная последовательность была сегментирована на файлы последовательностей по 50 т.п.о. Каждый отдельный 50 т.п.о. сегмент и повторяющиеся последовательности были визуализированы с помощью программы Dotter (см., например, Sonnhammer, E.L. Durbin, R. «A dot matrix program with dynamic threshold control suited for genomic DNA and protein sequence analysis», Gene 167: GC1-GC10 (1995)). Области повторяющейся последовательности были проанализированы в высоком разрешении в программе Dotter и приближенное изображение профиля локуса CRISPR дополнительно изучали, чтобы определить, присутствует ли последовательность повтора. Потенциальные локусы CRISPR были аннотированы вручную путем поиска текстового файла последовательности для несмежных повторов. Для каждого локуса-кандидата CRISPR повторы-кандидаты были дополнительно выровнены при помощи CLUSTALW и проанализированы с использованием WebLogo. После идентификации локуса CRISPR и его повторов были определены спейсеры как последовательности, расположенные между двумя последовательными повторами.
Как описано ниже, в общей сложности идентифицировано восемь локусов CRISPR: локус CRISPR со связанными cas-генами, названный «Bala1», и набор из семи локусов CRISPR, вероятно связанных, названных «CRISPRo».
В одном варианте осуществления предполагается, что последовательности CRISPR генома B. lactis Bl-04, предложенные по настоящему изобретению, можно использовать для идентификации локусов, повторов и спейсеров CRISPR в других видах бифидобактерий и других родственных бактериях. Любой подходящий метод для анализа сходства последовательностей, известный в данной области, можно использовать применительно к последовательностям CRISPR, раскрытым в данном документе, чтобы анализировать бактериальные геномы и идентифицировать родственные последовательности CRISPR. Например, как известно в данной области, анализ можно проводить при помощи NCBI BLAST с базой данных геномов микроорганизмов и GenBank.
2. Локус Bala1
В некоторых вариантах осуществления настоящее изобретение относится к композициям (например, выделенным нуклеиновым кислотам и векторам), способам и наборам, включающим нуклеотидные последовательности локуса Bala1 из генома B. lactis Bl-04.
Локус Bala1 содержит 2 kb нуклеотидную последовательность (SEQ ID NO: 1), изображенную на фигуре 1. Как показано на фигурах 2 и 3, локус Bala1 содержит набор из 23 повторов по 36 п.о. и 22 уникальных спейсеров с номерами S1-S22, длина которых варьируется от 34 п.о. до 39 п.о.
Локус CRISPR Bala1 из различных видов бифидобактерий, включая тип штамма B. animalis подвида animalis, подвергали амплификации методом ПЦР. Этот локус присутствовал исключительно в штаммах B. animalis подвида lactis, что указывало на его специфичность для подвидов.
Локус CRISPR Bala1 представляет собой четвертое семейство CRISPR, идентифицированное в бифидобактериях. Другими бифидобактериями с идентифицированными локусами CRISPR являются Blon1 (B. longum), Lhel1 (B. adolescentis) и Ldbu1 (B. catenulatum) (см. Horvath et al., J. Bacteriol., 2008, выше). Сравнение структуры генома B. lactis Bl-04, окружающей локусы CRISPR, с семью другими геномами бифидобактерий представлено на фигуре 4. Bala1 отличается от других локусов CRISPR, ранее идентифицированных в бифидобактериях, как с точки зрения области «повтор-спейсер» CRISPR (представленной на фигуре 3), так и присутствия шести расположенных ниже cas-генов.
Как описано в различных вариантах осуществления, раскрытых в данном документе, последовательности локуса Bala1 можно использовать в различных способах применения, включая, но не ограничиваясь ими: типирование штаммов, отслеживание штаммов, маркировку штаммов при помощи фагов и генетическое изменение устойчивости штаммов против вносимых нуклеиновых кислот.
Кроме того, в некоторых вариантах осуществления изобретения последовательности локуса Bala1 можно использовать в способах типирования, обнаружения, отслеживания и/или маркировки штаммов, описанных более подробно ниже.
3. Повторы Bala1
В локусе CRISPR Bala1 (SEQ ID NO: 1) находится 23 повтора по 36 п.о. каждый. Эти последовательности повторов представлены полужирными прописными буквами в изображении последовательности локуса на фигуре 1. 23 последовательности повторов Bala1 (R1-R23) изображены отдельно на фигуре 2.
Консенсусная нуклеотидная последовательность повтора Bala1 длиной 36 п. о.:
ATCTCCGAAGTCTCGGCTTCGGAGCTTCATTGAGGG (SEQ ID NO: 2). Эта консенсусная последовательность повтора сохраняется в первом 21 повторе (R1-R21). Однако в каждом из последних двух повторов (R22 и R23) имеются SNP (замена А на G) на 3′-конце, положение 36 в SEQ ID NO: 2. В повторе R3 6 также присутствуют замены С на Т в положениях 12 и 14 в SEQ ID NO: 2. Результаты анализа консенсусных последовательностей повторов длиной 36 п.о. с помощью программы WebLogo Университета Калифорнии продемонстрировали высокую степень консервативности последовательности (данные не представлены). Наблюдались незначительные вариации в повторе Bala1 в положениях 12 (замена Т на C), 14 (замена Т на С) и 36 (замена А на G).
В одном варианте осуществления настоящее изобретение относится к композиции выделенной нуклеиновой кислоты, содержащей последовательность повтора Bala1 (SEQ ID NO: 2). В другом варианте осуществления настоящее изобретение относится к композиции выделенной нуклеиновой кислоты, содержащей вариант последовательности повтора Bala1 (SEQ ID NO: 2), в котором вариация включает одну или более замен, выбранных из группы, состоящей из замены T в положении 12, замены T в положении 14 и замены A в положении 36.
4. Спейсеры Bala1
22 идентифицированных спейсера Bala1 соответствуют записанным строчными буквами последовательностям, расположенным между каждой парой повторов.
Локус Bala1 (SEQ ID NO: 1) включает 22 спейсера длиной от 34 до 39 п.о. Каждый из 22 спейсеров Bala1 расположен между парой повторов Bala1. Как изображено на фигуре 2, последовательность спейсера соответствует строчным буквам, расположенным после каждого набора из прописных букв, который соответствует последовательности повтора длиной 36 п.о.
Консенсусный анализ с помощью WebLogo последовательностей 22 спейсеров Bala1 продемонстрировал низкую степень консервативности, указывающую на относительную самобытность каждой последовательности. Среди 22 последовательностей спейсеров Bala1 был обнаружен небольшой, если вообще был обнаружен, консенсус. Таким образом, каждая последовательность спейсера Bala1 является высоко индивидуальной, в отличие от повторов или некоторых из спейсеров локуса CRISPRo.
В таблице 1 (ниже) представлен сводный перечень последовательностей 22 спейсеров Bala1, приведенных на фигуре 2.

Локус CRISPR Bala1 идентифицирован в геноме как Bl-04, так и DSM10140. Как показано на фигуре 3, наблюдался полиморфизм с точки зрения состава области «повтор-спейсер». В частности, хотя состав был одинаковым как в лидерном, так и в трейлерном конце локуса CRISPR, три последовательные внутренние единицы «повтор-спейсер» были уникальны для Bl-04.
Локус CRISPR Bala1 секвенировали для трех дополнительных штаммов B. lactis (Bi-07, B420 и HN019). Как показано на фигуре 3, сравнительный анализ локуса CRISPR Bala1 пяти различных штаммов B. lactis (Bl-04, Bi-07, DSM10140, B420 и HN019) выявил, что только Bl-04 и Bi-07 содержат 23 повтора и 22 спейсера. Геномы штаммов B. lactis DSM10140, B420 и HN019 не содержат спейсеров S10 (SEQ ID NO: 12), S11 (SEQ ID NO: 13) и S12 (SEQ ID NO: 14). Присутствие этих трех дополнительных областей «повтор-спейсер» также подтверждено сравнением при помощи графика Dotter областей «повтор-спейсер» в Bl-04 и DSM10140, который показал наличие делеции в три единицы между ними двумя.
Сходство с известными последовательностями фагов и с метагеномными последовательностями было выявлено для некоторых из спейсеров CRISPR Bala1. S3 имеет гомологию с фагом phi-BT1 AJ550940 Streptomyces и розеовирусом NZ_AAMV01000012.1. S17 имеет гомологию с вирусом лягушек AY548484.1. S19 имеет гомологию с метагеномом кишечника человека BABA01032251.1. S20 имеет гомологию с фаговым капсидным белком в Chromohalobacter salexigens CP000285.1 и с морским метагеномом AACY021620797.1. Кроме того, S8 имеет гомологию с метагеномом кишечника человека BABC01001407.1.
Интересно, что содержание GC локуса CRISPR Bala1 составляло примерно 49,74%, тогда как таковое в геноме составляет 60,19%, из чего можно предположить, что он мог быть приобретен со стороны от микроорганизма с низким содержанием GC, как уже обсуждалось ранее для локусов CRISPR (см. Godde and Bickerton, 2006; Horvath et al., «Comparative analysis of CRISPR loci in lactic acid bacteria genomes», Int. J. Food Microbiol., электронная публикация 15 июля 2008 г.).
В некоторых вариантах осуществления настоящее изобретение относится к композиции выделенной нуклеиновой кислоты, содержащей спейсер Bala1, обладающий последовательностью, выбранной из группы, состоящей из SEQ ID NO: 3-24.
В некоторых вариантах осуществления настоящее изобретение относится к выделенной нуклеиновой кислоте, содержащей последовательность повтора Bala1, связанную с последовательностью спейсера Bala1 в ориентации от 5'- к 3'-концу, где последовательность повтора Bala1 представляет собой SEQ ID NO: 2 или ее вариантную последовательность, и где последовательность спейсера Bala1 выбирают из группы, состоящей из SEQ ID NO: 3-24. В одном варианте осуществления вариант последовательности повтора Bala1 (SEQ ID NO: 2) содержит одну или более замен, выбранных из группы, состоящей из замены T в положении 12, замены T в положении 14 и замены A в положении 36.
5. Связанные с Bala1 cas-гены
Как правило, четыре cas-гена, носящие названия от cas1 до cas4, расположены в непосредственной близости от локуса CRISPR. Наиболее обычное расположение этих генов cas3-cas4-cas1-cas2. Хотя не все гены cas1-4 связаны со всеми локусами CRISPR, все они найдены в нескольких подтипах. Было предсказано, что белок Cas3 представляет собой HD-нуклеазу, слитую с DEAD-бокс геликазой, и, предположительно, помогает белкам каскада в ингибировании пролиферации вируса (см., например, Brouns et al. (2008) выше). Cas4 напоминает семейство RecB экзонуклеаз и содержит богатый цистеином мотив, указывающий на связывание ДНК. Было предсказано, что Cas1 является интегразой (см. Brouns et al. (2008) выше; Makarova et al., Biol. Direct 1: e60 (2005)). Cas1 является, как правило, сильно основным и единственным Cas-белком, систематически обнаруживаемым у всех видов, которые содержат локусы CRISPR. Было предсказано, что Cas2 является геном эндорибонуклеазы (см. Brouns et al. (2008) выше; Beloglazova et al., J. Biol. Chem. 283: 20361 (2008)).
Как показывает схематичное изображение локуса Bala1 на фигуре 3, шесть cas-генов расположены ниже от области «повтор-спейсер», включая универсальную нуклеазу cas1 (COG1518, TIGR00287), эндонуклеазу cas2 (COG1343, TIGR01573) и геликазу cas3 (COG1203, TIGR02621). Три дополнительных CRISPR-связанных белков семейства Csb (Csb1, Csb2 и Csb3) были также обнаружены в зонах, соседних с генами cas2 и cas3, как показано на фигуре 3. Аминокислотные последовательности, кодируемые тремя Cas-ферментами Bala1 и три белка Csb представлены на фигуре 5 (SEQ ID NO: 155-160).
Эти три не определенные cas-гены ранее не были охарактеризованы, в том числе в других локусах CRISPR бифидобактерий (см. Horvath et al., 2008 IJFM), но содержат консервативные элементы cas-типа (например, cas_GSU0053 и cas_GSU0054). Эти шесть cas-генов, вероятно, вовлечены в устойчивость к фагам, придаваемую последовательностями спейсеров и повторов Bala1. Шесть cas-генов локуса CRISPR Bala1 не имеют известных гомологов в геноме B. longum (см. фигуру 4).
В некоторых вариантах осуществления изобретение относится к выделенной нуклеиновой кислоте, кодирующей cas-ген локуса CRISPR Bala1, кодирующего аминокислотную последовательность Cas-белка, представленную на фигуре 5. Таким образом, аминокислотные последовательности можно выбирать из группы, состоящей из SEQ ID NO: 155, 156, 157, 158, 159 и 160. В одном варианте осуществления выбранная аминокислотная последовательность представляет собой любую из трех Cas-белков с SEQ ID NO: 156, 158 и 159.
В некоторых вариантах осуществления изобретение относится к выделенной нуклеиновой кислоте, способной кодировать полный комплект из шести cas-генов Bala1. Таким образом, выделенная нуклеиновая кислота может содержать нуклеотидные последовательности двух или более cas-генов, кодирующих две или более аминокислотных последовательностей, выбранных из группы, состоящей из SEQ ID NO: 155, 156, 157, 158, 159 и 160. В одном варианте осуществления выделенная нуклеиновая кислота содержит все шесть cas-генов локуса CRISPR Bala1.
6. Локусы CRISPRo
Были определены гомологии между типичными повторами в каждом идентифицированном локусе CRISPR в геноме Bl-04. Гомология между повторами в семи локусах CRISPR была достаточно высокой, чтобы определить их как подгруппу локусов «CRISPRo». Исходя из их вырожденности, похоже, что семь локусов CRISPRo происходят от одного предка CRISPR.
В некоторых вариантах осуществления настоящее изобретение относится к композициям (например, выделенных нуклеиновых кислот или векторов), способам и наборам, включающим нуклеотидные последовательности семи локусов CRISPRo из генома B. lactis Bl-04.
Локусы CRISPRo не содержат cas-генов. В некоторых вариантах осуществления изобретения каждый из семи локусов CRISPRo можно использовать для способов типирования, обнаружения, отслеживания и/или маркировки, как описано более подробно ниже.
Семь локусов CRISPRo названы по месту их расположения в геноме Bl-04. В частности, число в конце обозначения указывает на приблизительное местоположение в размерности килобаз (kb). Следовательно, семь локусов CRISPRo представляют собой CRISPRo9a, CRISPRo9b, CRISPRo91, CRISPRo164, CRISPRo228, CRISPRo245 и CRISPRo327.
Нуклеотидные последовательности семи локусов CRISPRo с их выровненными подпоследовательностями повторов и спейсеров изображены на фигурах 6-12. Идентификаторы последовательностей локусов CRISPRo являются следующими: CRISPRo9a (SEQ ID NO: 25), CRISPRo9b (SEQ ID NO: 26), CRISPRo91 (SEQ ID NO: 27), CRISPRo164 (SEQ ID NO: 28), CRISPRo228 (SEQ ID NO: 29), CRISPRo245 (SEQ ID NO: 30) и CRISPRo327 (SEQ ID NO: 31).
Каждый из семи локусов CRISPRo имеет от 8 до 11 последовательностей повторов и от 7 до 10 соответствующих последовательностей спейсеров. Нуклеотидные последовательности повторов и спейсеров, обнаруженные в семи локусах CRISPRo, приведены ниже в таблице 2.

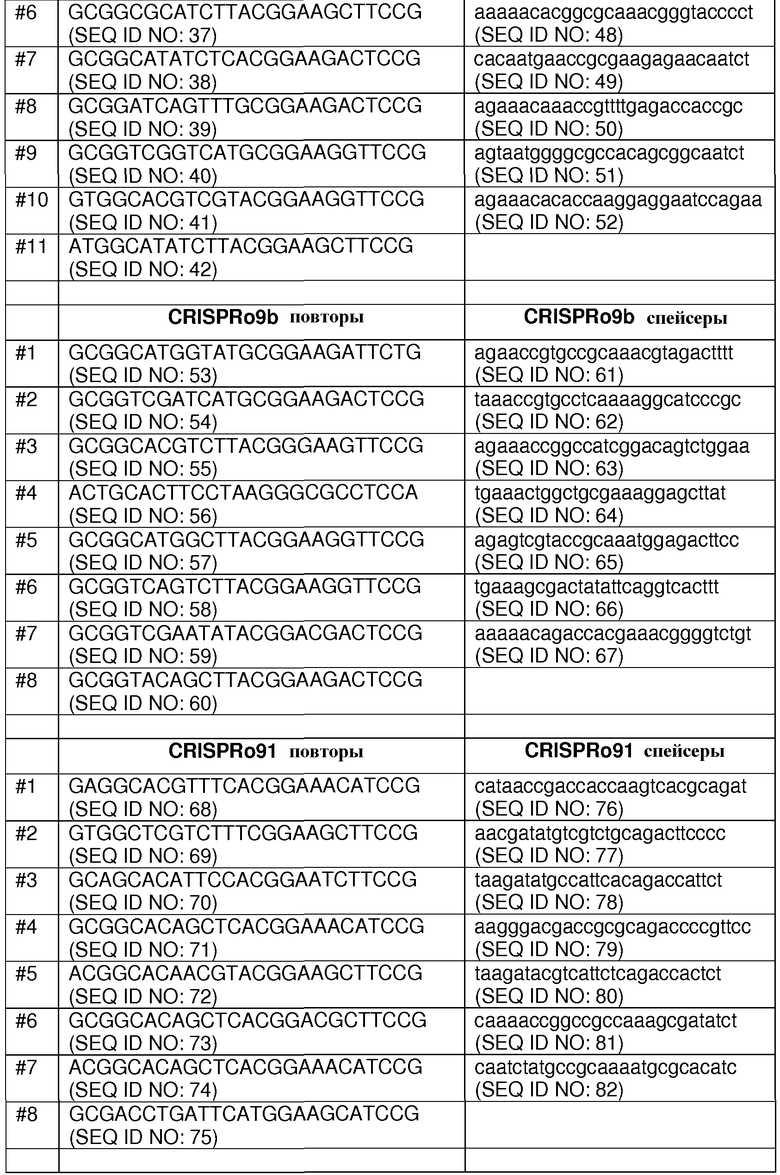
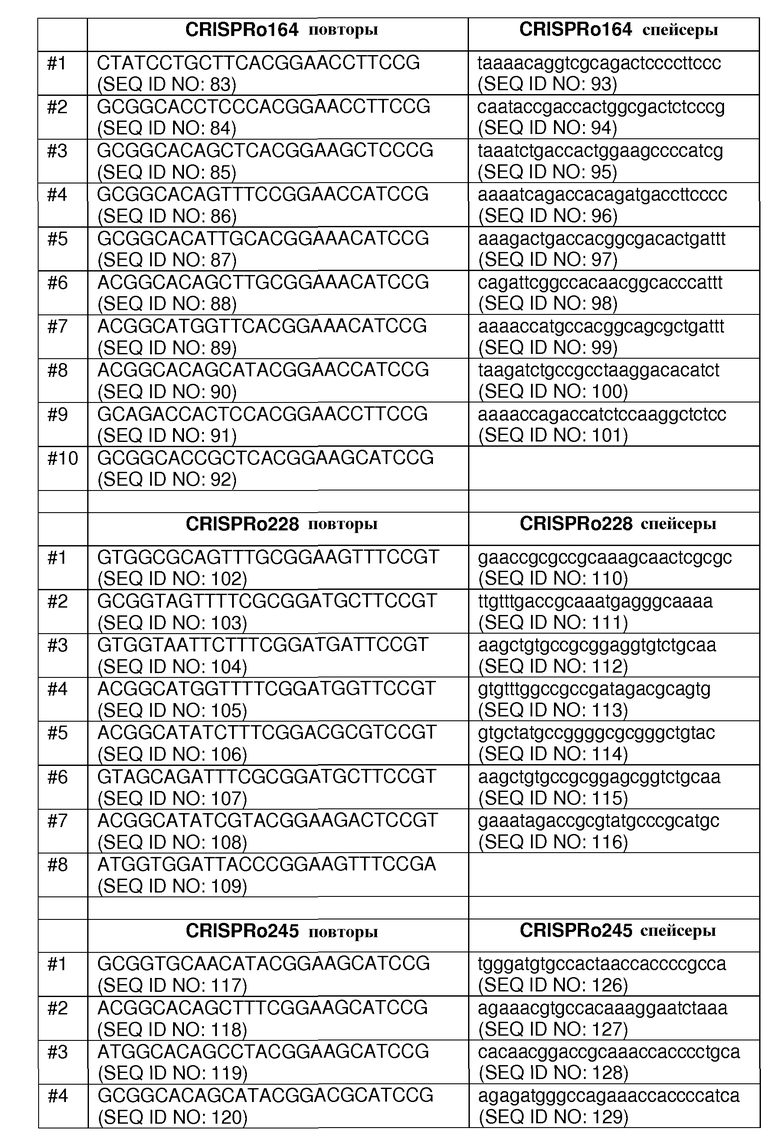

Как правило, все повторы CRISPRo имеют длину 25 п.о. (за исключением CRISPRo, длина которого 26 п.о.) и внутри каждого из семи локусов CRISPRo повторы обладают высокой степенью консенсуса последовательностей среди повторов, но не такой высокой как у повторов Bala1. В целом, все спейсеры CRISPRo имеют длину 26 или 27 п.о. Анализ с помощью WebLogo показал (как и для спейсеров Bala1), что среди последовательностей спейсеров CRISPRo, обнаруженных в каждом из семи локусов, консенсус невелик.
Выполняли консенсусный анализ последовательностей повторов и спейсеров CRISPRo, в котором сравнения проводили во всех семи локусах CRISPRo (данные не представлены). Установлено, что повторы CRISPRo имеют высокую степень гомологии по меньшей мере в 10 из 25 нуклеотидов (например, положения 3-4, 14-17 и 22-25) во всех 7 локусах. Напротив, спейсеры CRISPRo обладали высокой степенью гомологии только примерно в 2 из 27 нуклеотидов (например, положения 10-11) во всех 7 локусах.
Консенсусный анализ последовательностей комбинированной последовательности «повтор-спейсер» CRISPRo во всех 7 локусах, за исключением последнего повтора в каждом локусе, проводили с помощью WebLogo (данные не представлены). Интересно, что мотив последовательности, включающий первые 31 п.о., который содержит последовательности повторов и часть последовательности спейсера, является палиндромным. Считается, что палиндромный характер этой комбинированной последовательности может иметь решающее значение для функциональности локусов CRISPRo.
Таким образом, в другом варианте осуществления настоящее изобретение относится к выделенной нуклеиновой кислоте, содержащей последовательность повтора CRISPRo, связанную с последовательностью спейсера CRISPRo в ориентации от 5'- к 3'-концу, где последовательность повтора CRISPRo выбирают из группы, состоящей из SEQ ID NO: 32-42, 53-60, 68-75, 83-92, 102-109, 117-125 и 134-143, и где последовательность спейсера CRISPRo выбирают из группы, состоящей из SEQ ID NO: 43-52, 61-67, 76-82, 93-101, 110-116, 126-133 и 144-152.
B. Инженерия измененной устойчивости к нуклеиновым кислотам при помощи последовательностей CRISPR рекомбинантной B. lactis
Показано, что локусы CRISPR обеспечивают устойчивость против вносимых нуклеиновых кислот у прокариот. Например, показано, что специфические последовательности спейсеров в локусе CRISPR придают или изменяют устойчивость в штаммах S. thermophilus против конкретного фага (см., например, Barrangou et al., 2007 выше). Описанные общие методы инженерии измененной устойчивости к фагу в штаммах S. thermophilus можно использовать для инженерии измененной устойчивости в других бактериальных штаммах, обладающих локусами CRISPR.
Способы изменения бактериальной устойчивости к экзогенным нуклеиновым кислотам (например, фагам) путем добавления, удаления и/или модифицирования в эндогенных локусах CRISPR, имеющихся в бактериях, также раскрыты, например, в Russell & Klaenhammer, Appl. Environ. Microbiol. 67, 4361 (2001); и PCT публикации № WO2007/025097 A2, опубликованной 1 марта 2007 г., которая включена в данный документ посредством ссылки. Эти способы можно использовать в вариантах осуществления настоящего изобретения.
Как правило, минимальной геномной структурой, необходимой для придания связанной с CRISPR устойчивости против целевой нуклеиновой кислоты (или продукта ее экспрессии), является по меньшей мере один cas-ген (или один Cas-белок) и по меньшей мере два повтора, фланкирующие спейсер, CRISPR. Таким образом, в некоторых вариантах осуществления настоящее изобретение относится к способу изменения (например, придания или увеличения) устойчивости клетки против целевой нуклеиновой кислоты или продукта ее транскрипции, включающему (a) получение нуклеиновой кислоты, содержащей по меньшей мере один cas-ген и по меньшей мере два повтора CRISPR вместе со спейсером CRISPR, где последовательность спейсера CRISPR гомологична (например, как правило, на 100% идентична) последовательности целевой нуклеиновой кислоты (например, консервативной последовательности, необходимой для функционирования или выживания организма); и (b) трансформирование клетки указанной нуклеиновой кислотой, в результате чего трансформированная клетка становится устойчивой к указанной целевой нуклеиновой кислоте или продукту ее транскрипции. В некоторых вариантах осуществления способ дополнительно включает (c) создание контакта трансформированной бактерии с целевой нуклеиновой кислотой; и (d) выделение трансформированной бактерии, проявляющей измененную устойчивость к целевой нуклеиновой кислоте.
Способы трансформирования бактериальных клеток нуклеиновыми кислотами, содержащими последовательности CRISPR, таким образом, что трансформация приводит к встраиванию в геном посредством, например, гомологичной рекомбинации, хорошо известны в данной области, см., например, Sambrook et al. (Molecular Cloning: A Laboratory Manual, 2е издание, 1989 г., Cold Spring Harbor Laboratory Press) и Ausubel et al., Current Protocols in Molecular Biology (1995), John Wiley & Sons, Inc. Трансформированные клетки можно культивировать в хорошо известных условиях для репликации и/или экспрессии интегрированной последовательности нуклеиновой кислоты в бактерии.
В некоторых вариантах осуществления способа предварительно идентифицирован спейсер CRISPR, связанный с устойчивостью организма к целевой нуклеиновой кислоте или продукту ее транскрипции. В такой ситуации способ можно осуществлять, используя известную последовательность спейсера. В некоторых вариантах осуществления настоящего изобретения последовательность спейсера выбирают из группы спейсеров Bala1 B. lactis, состоящей из SEQ ID NO: 3-24.
У прокариот, геном которых уже содержит локус CRISPR и связанные cas-гены, связанную с CRISPR устойчивость можно изменять, модифицируя существующие последовательности CRISPR. В вариантах осуществления, в которых последовательность связанного с устойчивостью спейсера CRISPR уже известна, последовательность существующего спейсера CRISPR в организме можно модифицировать таким образом, что она становится гомологичной или идентичной связанному с устойчивостью спейсеру CRISPR. Альтернативно, единицу «повтор-спейсер», содержащую последовательность связанного с устойчивостью спейсера CRISPR, можно встраивать в существующий локус CRISPR, изменяя тем самым устойчивость организма. В других вариантах осуществления, в которых желательно уменьшить устойчивость, известный связанный с устойчивостью спейсер CRISPR можно модифицировать или удалять (как единицу «повтор-спейсер») из существующего локуса CRISPR, тем самым уменьшая или полностью ликвидируя устойчивость организма к целевой нуклеиновой кислоте.
Локус Bala1 B. lactis содержит типичные элементы CRISPR, необходимые для устойчивости к фагам, включая связанные cas-гены, консервативные повторы и последовательности спейсеров, которые обладают гомологией с известными фаговыми последовательностями. Например, нуклеотиды 18-36 спейсера S18 Bala1 (SEQ ID NO: 20) обладают 100% идентичностью с частью последовательности гена (ABE58753.1; Gl: 91796614), обнаруженного в геноме фага C. salexigens.
Анализ гомологии, проведенный для последовательностей спейсеров CRISPRo, выявил по меньшей мере один спейсер в каждом из семи локусов CRISPRo, который обладал значительной гомологией с известной фаговой последовательностью.

Основываясь на гомологии с фаговой последовательностью спейсеров CRISPRo, представленных в таблице 3, в некоторых вариантах осуществления настоящего изобретения эти гомологичные последовательности спейсеров можно использовать в способах инженерии измененной устойчивости к фагам в бактериальных штаммах, описанных в данном документе.
Последовательности локусов CRISPR B. lactis, раскрытые в данном документе, являются источником последовательностей повторов и спейсеров, которые можно использовать в способах инженерии измененной устойчивости в других бактериальных штаммах. Кроме того, информация о локусах Bala1 и CRISPRo обеспечивают необходимую «платформу» локуса CRISPR для инженерии B. lactis и других бифидобактерий. Например, спейсеры CRISPR из других организмов с известными характеристиками CRISPR-связанной устойчивости можно встраивать или иным образом генетически изменять в локусах Bala1 или CRISPRo B. lactis.
В одном варианте осуществления последовательности локуса Bala1 B. lactis и его связанные cas-гены можно использовать согласно вышеописанным способам для изменения устойчивости в штамме B. lactis против вносимых нуклеиновых кислот, в частности, для защиты от атак фага. В некоторых вариантах осуществления измененную устойчивость к фагу создают в бактериях с локусом CRISPR путем введения (то есть, встраивания при помощи методов рекомбинантной ДНК) спейсера Bala1 или CRISPRo в локус CRISPR бактериального генома, где спейсер Bala1 или CRISPRo имеет последовательность, выбранную из группы, состоящей из SEQ ID NO: 3-24, 43-52, 61-67, 76-82, 93-101, 110-116, 126-133 и 144-152.
В некоторых вариантах осуществления различных способов, раскрытых в данном документе, измененную устойчивость к фагу создают в бактериальном штамме с локусом CRISPR путем введения единицы «повтор-спейсер» Bala1 или CRISPRo в локус CRISPR бактериального генома. В таком варианте осуществления единица «повтор-спейсер» Bala1 содержит последовательность повтора, выбранную из SEQ ID NO: 2 и ее вариантов, и последовательность спейсера, выбранную из SEQ ID NO: 3-24; единица «повтор-спейсер» CRISPRo содержит последовательность повтора, выбранную из SEQ ID NO: 32-42, 53-60, 68-75, 83-92, 102-109, 117-125 и 134-143, и последовательность спейсера, выбранную из SEQ ID NO: 43-52, 61-67, 76-82, 93-101, 110-116, 126-133 и 144-152.
В некоторых вариантах осуществления описанных выше способов последовательность спейсера CRISPR B. lactis, введенная в существующий локус CRISPR бактерии, содержит последовательность, обладающую по меньшей мере 98% идентичностью, по меньшей мере 99% идентичностью или по меньшей мере 100% идентичностью с последовательностью целевой нуклеиновой кислоты фага, к которому нужна устойчивость. В некоторых вариантах осуществления целевая нуклеиновая кислота консервативна в фаге и в некоторых вариантах осуществления содержит последовательность, необходимую для выживания фага.
В различных вариантах осуществления способов изменения устойчивости в клетках, раскрытых в данном документе, инженерия локуса CRISPR в клетке, для которой необходима измененная устойчивость, может включать, но не ограничивается ими, добавление (например, вставку), делетирование (например, удаление) или изменение (например, мутацию) последовательности спейсеров CRISPR в клетке таким образом, что локус CRISPR клетки начинает обладать гомологией (например, повышенной гомологией после инженерии) по меньшей мере с одним спейсером CRISPR организма с известной CRISPR-связанной устойчивостью (например, клетка B. lactis). Такая инженерия может приводить к созданию клетки, которая была в значительной степени чувствительна к целевой нуклеиновой кислоте или продукту ее транскрипции, но становится в значительной степени устойчивой к целевой нуклеиновой кислоте или продукту ее транскрипции.
В некоторых вариантах осуществления изобретение относится к способу изменения устойчивости клетки против целевой нуклеиновой кислоты, где клетка содержит локус CRISPR и связанные cas-гены, включающему (a) идентификацию одного или более связанных с устойчивостью спейсеров CRISPR в организме, устойчивом к целевой нуклеиновой кислоте; и (b) модификацию последовательности по меньшей мере одного спейсера в локусе CRISPR клетки таким образом, что его последовательность становится гомологичной или идентичной последовательности связанного с устойчивостью спейсера CRISPR. В вариантах осуществления настоящего изобретения применяют способ, в котором организм, устойчивый к целевой нуклеиновой кислоте, представляет собой бифидобактерии (например, B. lactis), а связанные с устойчивостью спейсеры CRISPR выбирают из спейсеров Bala1 и CRISPRo с SEQ ID NO: 3-24, 43-52, 61-67, 76-82, 93-101, 110-116, 126-133 и 144-152.
В некоторых вариантах осуществления предложены способы уменьшения или снижения устойчивости клетки, содержащей по меньшей мере один или более cas-генов или белков и один или более, предпочтительно два или более, повторов CRISPR, против целевой нуклеиновой кислоты или продукта ее транскрипции. Согласно данному варианту осуществления способ включает этапы (a) идентификации одного или более спейсеров CRISPR в организме, который в значительной степени устойчив к целевой нуклеиновой кислоте или продукту ее транскрипции; и (b) изменение последовательности одного или более спейсеров CRISPR в клетке таким образом, что спейсер(ы) CRISPR начинают обладать меньшей степенью гомологии со спейсером(ами) CRISPR в организме.
Различные варианты осуществления способов инженерии измененной устойчивости к целевой нуклеиновой кислоте, раскрытых в данном документе, можно использовать в целом ряде применений: (i) инженерия устойчивости к фагам; (ii) инженерия устойчивости к переносу плазмид; (iii) инженерия устойчивости к мобильным генетическим элементам; (iv) инженерия устойчивости к генам устойчивости к антибиотикам; (v) инженерия устойчивости к генам, кодирующим факторы вирулентности, и (vii) инженерия устойчивости к новым последовательностям. Различные последовательности локусов CRISPR B. lactis и связанные последовательности повторов и спейсеров, раскрытые в данном документе, обеспечивают платформу для инженерии измененной устойчивости в B. lactis и родственных бифидобактериях с родственными локусами CRISPR.
Как правило, в вариантах осуществления для изменения устойчивости к фагу конкретные спейсеры CRISPR, происходящие из ДНК бактериофага, добавляют в локус CRISPR бактериальной клетки таким образом, чтобы обеспечить устойчивость против данного конкретного бактериофага и предотвратить атаку фага. Кроме того, мишенью могут становиться конкретные области внутри генома фага (белки специфичности к хозяину), которые обеспечивают особое узнавание фаг-хозяин или которые высоко консервативны в ДНК фага, такие как последовательности из генов геликазы или праймазы, структурные белки головы и хвоста или белки с консервативными доменами (например, геликаза, перфорирование, лизин и другие) или консервативные последовательности среди важных генов фага. Таким образом, информацию о конкретных последовательностях фага можно использовать для модификации существующих последовательностей спейсеров CRISPR (например, спейсеров, обнаруженных в локусах Bala1 и CRISPRo) для изменения устойчивости к конкретному фагу.
В некоторых вариантах осуществления инженерию измененной устойчивости к переносу плазмиды в бактериальном штамме можно осуществлять способами, раскрытыми в данном документе. Конкретные спейсеры CRISPR, происходящие из плазмидной ДНК, добавляют в существующий локус CRISPR таким образом, чтобы обеспечить устойчивость против данной конкретной плазмиды, тем самым предотвращая перенос чужеродной ДНК в клетки штамма. В некоторых вариантах осуществления конкретные области в целевой плазмидной ДНК, такие как последовательности в составе точки начала репликации плазмиды, выбирают для внесения в локус CRISPR таким образом, чтобы обеспечить иммунитет против плазмидной ДНК.
В некоторых вариантах осуществления способы изменения устойчивости к мобильному генетическому элементу осуществляют аналогичным образом, добавляя спейсеры CRISPR, происходящие из ДНК мобильного генетического элемента, в существующий локус CRISPR бактериального штамма таким образом, чтобы обеспечить устойчивость против мобильных генетических элементов, таких как перемещающиеся элементы и инсерционные последовательности. Такая генетически измененная устойчивость может предотвращать перенос чужеродной ДНК и генетический дрейф в генно-инженерном бактериальном штамме. В частности, мишенью могут становиться конкретные области внутри транспозонов и инсерционных последовательностей, чтобы обеспечить иммунитет против мобильных генетических элементов. Например, мишени могут включать конъюгативные транспозоны (Tn97<5), транспозоны класса II (Tn501) или инсерционные последовательности (IS2d).
В некоторых вариантах осуществления способы изменения устойчивости к генам устойчивости к антибиотикам осуществляют, добавляя спейсеры CRISPR, происходящие из генов, кодирующих устойчивость к антибиотикам, в существующий локус CRISPR бактериального штамма. Такая измененная устойчивость может предотвращать перенос генов, придающих устойчивость к антибиотикам, в бактериального хозяина, тем самым уменьшая риск приобретения маркеров устойчивости к антибиотикам. Например, мишени могут включать vanR, ген, придающий устойчивость к ванкомицину, или tetR, ген, придающий устойчивость к тетрациклину, или может быть направленность на ингибиторы бета-лактамазы.
В некоторых вариантах осуществления последовательности спейсеров CRISPR, происходящие из генов, кодирующих факторы вирулентности, можно добавлять в локус CRISPR бактерий, чтобы обеспечить устойчивость против переноса генов, придающих вирулентность, в бактерию. Например, мишенью могут быть факторы, часто способствующие вирулентности патогенных микроорганизмов, такие как токсины, интерналины и гемолизины.
В некоторых вариантах осуществления новые последовательности спейсеров можно синтезировать de novo, конструировать и интегрировать в локус CRISPR в выбранном бактериальном хозяине, чтобы обеспечить устойчивость к конкретной идентичной и новой последовательности, присутствующей в инфекционной молекуле ДНК.
C. Обнаружение штаммов, типирование штаммов и/или отслеживание штаммов при помощи последовательностей CRISPR B. lactis
В некоторых вариантах осуществления настоящего изобретения последовательности локусов Bala1 и/или CRISPRo и части этих последовательностей (например, повторы, спейсеры и их сочетания) можно использовать для обнаружения, отслеживания и/или типирования бактерий, в которых они присутствуют. Способы обнаружения, типирования и отслеживания штаммов при помощи последовательностей CRISPR, которые можно использовать в вариантах осуществления настоящего изобретения, раскрыты, например, в опубликованной патентной заявке США 2006/01990190 A1, опубликованной 7 сентября 2006 г., которая включена в данный документ посредством ссылки.
Локус Bala1 и по меньшей мере один из семи локусов CRISPRo, раскрытых в данном документе, идентифицированы во всех штаммах B. animalis, протестированных до настоящего времени. Тем не менее, можно обнаружить различия между последовательностями локусов Bala1 и CRISPRo, обнаруженных в различных протестированных штаммах B. animalis. Таким образом, в некоторых вариантах осуществления настоящего изобретения наличие различий в данных последовательностях используют для обнаружения, типирования и/или отслеживания конкретных штаммов, различающихся по этим последовательностям.
В некоторых вариантах осуществления штамм обнаруживают и/или отличают от другого штамма (то есть, типируют) путем амплификации части последовательности локуса CRISPR в штамме, который был определен, чтобы его отличить. Методы амплификации специфических генетических последовательностей в бактериях хорошо известны в данной области (например, ПЦР, ЛЦР, изотермальные методы). Как правило, локус CRISPR или его часть из известной бактерии-источника (например, родительской бактерии) и из неизвестной бактерии амплифицируют и/или секвенируют любым подходящим методом, известным в данной области. Определенные последовательности и/или определенные ампликоны (например, в зависимости от размера) сравнивают для выяснения, являются ли они одинаковыми или различными.
В некоторых вариантах осуществления настоящего изобретения локус CRISPR или его часть из бактерии-источника (или известной) и из неизвестной бактерии (например, анализируемого образца) сравнивают путем амплификации локуса CRISPR или его части и сравнения характеристик ампликонов (например, анализа размеров ампликонов электрофорезом в агарозном геле). В других вариантах осуществления настоящего изобретения локус CRISPR или его часть из известной бактерии-источника и из неизвестной бактерии-источника сравнивают путем секвенирования локуса CRISPR или его части из каждой из них и сравнения последовательностей.
В некоторых вариантах осуществления настоящего изобретения обнаружение, типирование и/или отслеживание штаммов осуществляют способом, включающим (a) амплификацию геномной ДНК из представляющего интерес штамма при помощи по меньшей мере одной пары праймеров, где геномная ДНК содержит по меньшей мере часть последовательности локуса CRISPR; и (b) обнаружение ампликона, полученного на этапе (a), при этом указанный обнаруженный ампликон указывает на тип штамма. В некоторых вариантах осуществления каждый праймер из пары комплементарен по меньшей мере части последовательности локуса CRISPR.
Как правило, ПЦР используют для амплификации геномной ДНК, полученной из представляющего интерес штамма. Используя последовательности CRISPR, раскрытые в данном документе, или последовательности, найденные в опубликованных геномах бактерий, несложно определить отличающиеся последовательности CRISPR (например, которые отличаются по числу повторов или спейсеров) и разработать пары праймеров для ПЦР, которые можно использовать для амплификации областей, содержащих эти последовательности.
В некоторых вариантах осуществления бактерии сравнивают, амплифицируя, а затем секвенируя локус CRISPR или его часть. Например, как 5'-, так и 3'-концы локусов можно амплифицировать и/или секвенировать и сравнивать.
В некоторых вариантах осуществления сравнивают один конец (например, 5'-конец) локусов CRISPR. В других вариантах осуществления сравнивают по меньшей мере последний повтор CRISPR на 3'-конце локуса CRISPR и/или по меньшей мере последний спейсер CRISPR на 3'-конце локуса CRISPR и/или по меньшей мере первый повтор CRISPR на 5'-конце локуса CRISPR и/или по меньшей мере первый спейсер CRISPR (например, внутреннюю часть первого спейсера CRISPR) на 5'-конце локуса CRISPR. В некоторых вариантах осуществления сравнивают по меньшей мере первый повтор CRISPR на 5'-конце локуса CRISPR и/или по меньшей мере первый спейсер CRISPR (например, внутреннюю часть первого спейсера CRISPR) на 5'-конце локуса CRISPR. В некоторых дополнительных вариантах осуществления сравнивают по меньшей мере последний спейсер CRISPR (например, внутреннюю часть последнего спейсера CRISPR) на 3'-конце локуса CRISPR и/или по меньшей мере первый спейсер CRISPR (например, внутреннюю часть первого спейсера CRISPR) на 5'-конце локуса CRISPR. В некоторых дополнительных вариантах осуществления сравнивают по меньшей мере первый спейсер CRISPR (например, внутреннюю часть первого спейсера CRISPR) на 5'-концах локусов CRISPR.
В одном варианте осуществления обнаружения, типирования и/или отслеживания штаммов отличительной особенностью CRISPR является число единиц «повтор-спейсер» в локусе CRISPR. То есть, в одном штамме может быть 12 повторов и 11 спейсеров, а в другом штамме может быть только 8 повторов и семь спейсеров. Амплификация областей CRISPR в этих двух штаммах приведет к получению ампликонов ПЦР, сильно различающихся по размерам. Обнаружение данного ампликона и определение его размера и того, чем он отличается от другого штамма, является одним из вариантов осуществления для типирования штаммов.
Поскольку локусы CRISPR обычно изменяют путем добавления новых единиц «повтор-спейсер» к 5'-концу области «повтор-спейсер» локуса CRISPR, в некоторых вариантах осуществления способ обнаружения или типирования нового образованного штамма включает получение ампликона, включающего 3'-конец лидерной последовательности CRISPR. Таким образом, в некоторых вариантах осуществления способ типирования штаммов включает использование праймера, сконструированного для гибридизации с последовательностью, содержащей 3'-конец лидера CRISPR. В одном варианте осуществления праймер гибридизуется с последовательностью, содержащей 3'-конец лидера CRISPR и 5'-конец первого повтора.
В одном варианте осуществления изменения в штамме можно отслеживать путем амплификации образцов штамма с течением времени. Изменения в быстро меняющихся последовательностях CRISPR будут проявляться как изменения в размерах обнаруженного ампликона. Например, если новые пары повторов добавляются к 5'-концу области CRISPR, это приведет к увеличению размера наибольшего ампликона, обнаруженного в штамме.
В одном варианте осуществления данный способ обнаружения, типирования и/или отслеживания штаммов можно использовать для обнаружения антропогенных изменений в CRISPR, например, инженерии устойчивости к фагу путем введения нового спейсера в существующий локус CRISPR. Таким образом, предполагается, что способы обнаружения, типирования и/или отслеживания штаммов можно использовать для мониторинга вариантов осуществления маркировки штаммов, раскрытых в данном документе. Например, если штамм бифидобактерий маркирован спейсером CRISPR после воздействия фага, включение новой единицы «повтор-спейсер» можно контролировать способами обнаружения, типирования и/или отслеживания штаммов, описанными выше.
Как правило, в вариантах осуществления настоящего изобретения, касающихся обнаружения, типирования и отслеживания штаммов, получают ампликон ПЦР при помощи ампликонов, комплементарных области последовательностей локусов Bala1 или CRISPRo, имеющейся в геномных образцах из двух штаммов B. lactis. Обнаруженное различие в ампликонах из двух образцов (определенное на основании различий в последовательности и/или размере ампликонов) свидетельствует о разнице в числе единиц «повтор-спейсер», присутствующих в локусах CRISPR из двух штаммов. Если установлено, что два образца бифидобактерий являются идентичными, то можно сделать вывод о том, что они принадлежат к одному типу. Если два образца включают архивный образец штамма и образец, полученный после того, как штамм бифидобактерий прошел через множество делений в производственном процессе, сравнение между ними двумя выявляет, претерпел ли штамм изменения при воздействии фага или вследствие других воздействий. Если один образец представляет собой родительский штамм, а другой представляет собой тот же самый родительский штамм после воздействия фага, сравнение может подтвердить, произошла ли маркировка штамма спейсером CRISPR.
D. Маркировка штаммов при помощи последовательностей CRISPR B. lactis
В некоторых вариантах осуществления настоящее изобретение относится к способам маркировки бактериального штамма при помощи последовательностей локусов Bala1 и/или CRISPRo или частей таких последовательностей (например, повторов, спейсеров и их сочетаний). Способы маркировки штаммов последовательностями CRISPR, применимые в вариантах осуществления настоящего изобретения, раскрыты, например, в опубликованной заявке США 2008/0124725 A1, опубликованной 29 мая 2008 г., которая включена в данный документ посредством ссылки.
Как правило, в вариантах осуществления маркировки штаммов бактериальный штамм, который предстоит маркировать, подвергают воздействию фага и инфекция индуцирует добавление последовательности CRISPR (например, единицы «повтор-спейсер») к локусу CRISPR в бактериальном штамме. Эта добавленная единица «повтор-спейсер» действует как генетический маркер (то есть, маркерная последовательность) для бактериального штамма, который можно обнаруживать при помощи любого из хорошо известных методов, описанных выше в контексте типирования, обнаружения и отслеживания штаммов (например, ПЦР, секвенирование, гибридизация с иммобилизованным зондом).
Таким образом, в некоторых вариантах осуществления настоящее изобретение относится к способу маркировки штамма бифидобактерий, включающему воздействие на клетки соответствующего родительского штамма, содержащего по меньшей мере часть локуса CRISPR, по меньшей мере одной экзогенной последовательностью нуклеиновой кислоты, тем самым получая маркированную бактериальную клетку с локусом CRISPR, содержащим по меньшей мере одну дополнительную единицу «повтор-спейсер» по сравнению с клетками родительского штамма.
В некоторых вариантах осуществления настоящее изобретение относится к способам маркировки бактериального штамма, включающим (a) воздействие на родительский бактериальный штамм фагом; (b) отбор нечувствительного к фагу мутанта; (c) сравнение локуса CRISPR или его части из родительского штамма и нечувствительного к фагу мутантного штамма и (d) отбор маркированного бактериального штамма, содержащего дополнительную единицу «повтор-спейсер» в локусе CRISPR, которая отсутствует в родительском бактериальном штамме.
В альтернативных вариантах осуществления настоящее изобретение относится к способам маркировки локуса CRISPR бактериального штамма при помощи методов рекомбинантной ДНК, известных в данной области, а не вследствие воздействия фага. Например, в некоторых вариантах осуществления получают синтетические олигонуклеотиды и используют их для трансформации родительских бактерий с целью получения маркированных по CRISPR бактерий.
Последовательности локусов CRISPR B. lactis, раскрытые в данном документе, предоставляют источник последовательностей повторов и спейсеров, которые можно использовать в способах маркировки других бактериальных штаммов. Кроме того, последовательности локусов Bala1 и CRISPRo, раскрытые в данном документе, предоставляют целевые локусы CRISPR, которые можно маркировать спейсерами из других источников. Например, спейсеры CRISPR из других микроорганизмов можно встраивать или иным образом генетически вводить в локусы Bala1 или CRISPRo B. lactis. Таким образом, информация о последовательности локусов Bala1 или CRISPRo, раскрытая в данном документе, предоставляет данные для понимания того, был ли штамм маркирован.
Таким образом, в некоторых вариантах осуществления настоящее изобретение относится к маркированному бактериальному штамму, где штамм не является B. lactis и содержит локус CRISPR, включающий последовательность спейсера Bala1 или CRISPRo, выбранную из SEQ ID NO: 3-24, 43-52, 61-67, 76-82, 93-101, 110-116, 126-133 и 144-152.
Для инфицирования клеток фаг инъецирует или переносит свою нуклеиновую кислоту в клетку, при этом нуклеиновая кислота фага существует независимо от клеточного генома. В некоторых вариантах осуществления инфекция приводит к экспрессии (то есть, транскрипции и трансляции) нуклеиновой кислоты фага в клетке и продолжению жизненного цикла фага. Таким образом, в некоторых вариантах осуществления настоящего изобретения после воздействия фага бактериальный штамм не только маркируется, но также приобретает устойчивость к последующей инфекции и/или размножению фага по сравнению с соответствующим родительским бактериальным штаммом.
В некоторых вариантах осуществления маркированная бактерия является нечувствительной или в значительной степени нечувствительной к последующей инфекции и/или размножению фага. В дополнительных вариантах осуществления маркированная бактерия является нечувствительной или в значительной степени нечувствительной к одному или нескольким механизмам, которые использует фаг для инфицирования и/или размножения в бактерии. В следующих вариантах осуществления маркированная бактерия является нечувствительной или в значительной степени нечувствительной ко всем механизмам, которые использует фаг для инфицирования и/или размножения в бактерии. В дополнительных вариантах осуществления в маркированной бактерии развиваются один или несколько механизмов, которые ослабляют, инактивируют или уничтожают фаг в процессе инфекционного цикла. Таким образом, в некоторых вариантах осуществления настоящее изобретение относится к маркированным штаммам, выбранным при помощи стандартных методов скрининга, известных в данной области, для выделения нечувствительных к фагу мутантов.
В следующих вариантах осуществления изобретение относится к клеточным культурам, содержащим маркированные бактериальные штаммы, полученные описанными выше способами.
Манеру и способ осуществления настоящего изобретения специалисты в данной области смогут легче понять с помощью следующих далее примеров, данные примеры не предназначены каким-либо образом ограничивать сферу применения настоящего изобретения или прилагающейся формулы изобретения.
ПРИМЕРЫ
Следующие примеры приведены с целью продемонстрировать и дополнительно проиллюстрировать конкретные варианты осуществления и аспекты настоящего изобретения и не могут быть истолкованы как ограничивающие сферу его применения.
В следующем далее описании экспериментов используются следующие сокращения: °C (градусы Цельсия); H2O (вода); dH2O (деионизированная вода); HCl (соляная кислота); aa (аминокислота); п.о. (пара оснований); kb (пара килобаз); кД (kD) (килодальтоны); г (граммы); мкг (микрограммы); мг (миллиграммы); мкл (микролитры); мл (миллилитры); мм (миллиметры); мкм (микрометр); М (молярный); мМ (миллимолярный); мкМ (микромолярный); MW (молекулярный вес); с (секунды); мин (минута/минуты); ч (час/часы); NaCl (хлорид натрия); PBS (фосфатно-солевой буферный раствор [150 мМ NaCl, 10 мМ натрий-фосфатный буфер, pH 7,2]); ПЦР (полимеразная цепная реакция); SDS (додецилсульфат натрия); w/v (вес к объему); v/v (объем к объему); ATCC (Американская коллекция типовых культур, Rockville, MD); BD BioSciences (ранее CLONTECH Laboratories, Palo Alto, CA); Invitrogen (Invitrogen Corp., San Diego, CA); и Sigma (Sigma Chemical Co., St. Louis, MO).
ПРИМЕР 1
Обнаружение методом ПЦР локуса Bala1 B. lactis
Данный пример иллюстрирует, как можно использовать ПЦР-амплификацию для обнаружения локуса Bala1 в штамме B. lactis бифидобактерий и отличать этот штамм от других штаммов бифидобактерий, имеющих локус Bala1.
На основании последовательности локуса Bala1 B. lactis (SEQ ID NO: 1) была разработана пара праймеров для ПЦР.
Праймер 1 представляет собой 24-мерный олигонуклеотид с последовательностью: 5'-TGAGGGaagccgaactcaatcaca-3' (SEQ ID NO: 153). Праймер 1 комплементарен антисмысловой цепи 24 п.о. последовательности Bala1, охватывающей часть повтора R4 и часть соседнего спейсера S4 (см. таблицу 1 выше). Часть праймера 1, перекрывающаяся с повтором R4, представлена всеми прописными буквами.
Праймер 2 представляет собой 24-мерный олигонуклеотид с последовательностью: 5'-ttggatgcaagCCCTCAATGAAGC-3' (SEQ ID NO: 154). Праймер 2 комплементарен 24 п.о. последовательности Bala1, охватывающей часть повтора R16 и часть соседнего спейсера S16 (см. таблицу 1 выше). Часть праймера 2, перекрывающаяся с повтором R16, представлена всеми прописными буквами.
Пара из праймера 1 и праймера 2 разработана для получения ампликона, содержащего 12 единиц «повтор-спейсер» Bala1, включая все спейсеры от спейсера S4 до спейсера S15 включительно (см. фигуру 2 и таблицу 1). Размер полученного ампликона Bala1 по прогнозам будет составлять 874 п.о.
Пару из праймера 1 и праймера 2 использовали в реакции ПЦР с образцами из четырех различных штаммов B. lactis (штаммы: Bl-04, Bi-07, HN019 и B420) и четырех других видов бифидобактерий: B. bifidum (Bb-06), B. longum (Bl-05), B. infantis (Bi-08) и B. breve (Bb-03). Кроме того, был амплифицирован образец (Bb-02), содержащий смесь B. bifidum и B. lactis.
Образцы амплифицировали в стандартных условиях ПЦР для получения меченых ампликонов. Смесь для ПЦР (на 50 мкл реакционной смеси) содержала смесь Mastermix компании Eppendorf (20 мкл); воду (27 мкл); праймер 1 (прямой праймер) (1 мкл); праймер 2 (обратный праймер) (1 мкл); матричную ДНК (1 мкл). Первоначальную денатурацию проводили в течение 5 минут при 95°C. Термическое циклирование включало 30 циклов по 30 с денатурации при 95°C, 30 с отжига при 58°C и 1 мин достраивания при 72°C. Заключительное достраивание проводили в течение 5 минут при 72°C.
После амплификации реакционные смеси ПЦР анализировали электрофорезом в агарозном геле для определения относительных размеров меченых ампликонов.
Одна четкая полоса была обнаружена на дорожках агарозного геля, соответствующих образцам штаммов Bl-04 и Bi-07, что означало наличие ампликона длиной примерно 870 п.о., который был предсказан, исходя из последовательности Bala1 B. lactis (SEQ ID NO: 1) и дизайна праймеров 1 и 2. Напротив, на дорожках геля, содержащих штаммы B. lactis HN019 и B420, отсутствовала полоса 874 п.о., но вместо нее у каждого образца присутствовала одна четкая полоса, соответствующая ампликону ПЦР длиной примерно 650 п.о., которую предсказали, исходя из их последовательностей CRISPR, имеющих на три спейсера меньше. Таким образом, как и предсказано, наблюдаемые размеры ампликона из штаммов HN019 и B420 соответствуют 9 единицам «повтор-спейсер», в отличие от ампликонов, содержащих 12 единиц, в штаммах Bl-04 и Bi-07.
На дорожке агарозного геля, содержащей образец Bb02 (который представлял собой смесь B. lactis и B. bifidum Bl-04), также наблюдали одну четкую полосу, соответствующую ампликону длиной 874 п.о., как и ожидалось из-за присутствия в образце штамма Bl-04. Однако в результате реакций ПЦР с использованием образцов из четырех других видов бифидобактерий не было получено поддающихся обнаружению ампликонов. Это указывает на то, что последовательности любых локусов CRISPR, присутствующих в данных видах, достаточно сильно отличаются, чтобы исключить амплификацию с использованием данных праймеров.
Наблюдаемые различия в ампликонах, полученных из различных штаммов B. lactis, четко иллюстрируют возможность использования этого фундаментального основанного на амплификации способа для типирования, обнаружения и/или отслеживания штаммов.
Рядовой специалист в данной области признает, что этот метод также можно легко использовать в других вариантах осуществления изобретения, включая определение того, были ли конкретные единицы «повтор-спейсер» встроены в локус CRISPR штамма вследствие экспериментов по маркировке штамма или по изменению устойчивости к фагу, описанных в другом месте данного документа.
Специалисты в данной области легко признают, что настоящее изобретение хорошо приспособлено для выполнения задач и получения упомянутых, а также присущих ему конечных результатов и преимуществ. Композиции и способы, описанные в данном документе, являются репрезентативными, примерными вариантами осуществления и не предназначены для ограничения объема изобретения.
В то время как конкретные варианты осуществления настоящего изобретения были проиллюстрированы и описаны, специалистам в данной области будет очевидно, что можно применять различные другие изменения и модификации без отклонения от сущности и объема изобретения. Вследствие этого предусмотрено, что все такие изменения и модификации, которые находятся в рамках данного изобретения, будут охвачены прилагающейся формулой изобретения.
Изобретение, иллюстративно описанное в данном документе, надлежащим образом может осуществляться на практике в отсутствие любого элемента(ов) или ограничения(ий), конкретно не описанных в данном документе. Употребляемые термины и выражения используются в качестве средств описания, но не ограничения, и при использовании таких терминов и выражений не является целью исключение любых эквивалентов представленных и описанных характерных особенностей или их частей.
В данном документе изобретение описано в широком смысле и в общих чертах. Все более узкие варианты и менее общие группировки, попадающие под общее описание, также являются частью данного изобретения. Это включает общее описание изобретения с оговоркой или отрицательное ограничение, исключающее какой-либо предмет из класса объектов, независимо от того, изложен ли специально в данном документе вырезанный материал.
Все патенты и публикации включены в данный документ посредством ссылок в такой же степени, как если бы каждая отдельная публикация была специально и индивидуально включена посредством ссылки.


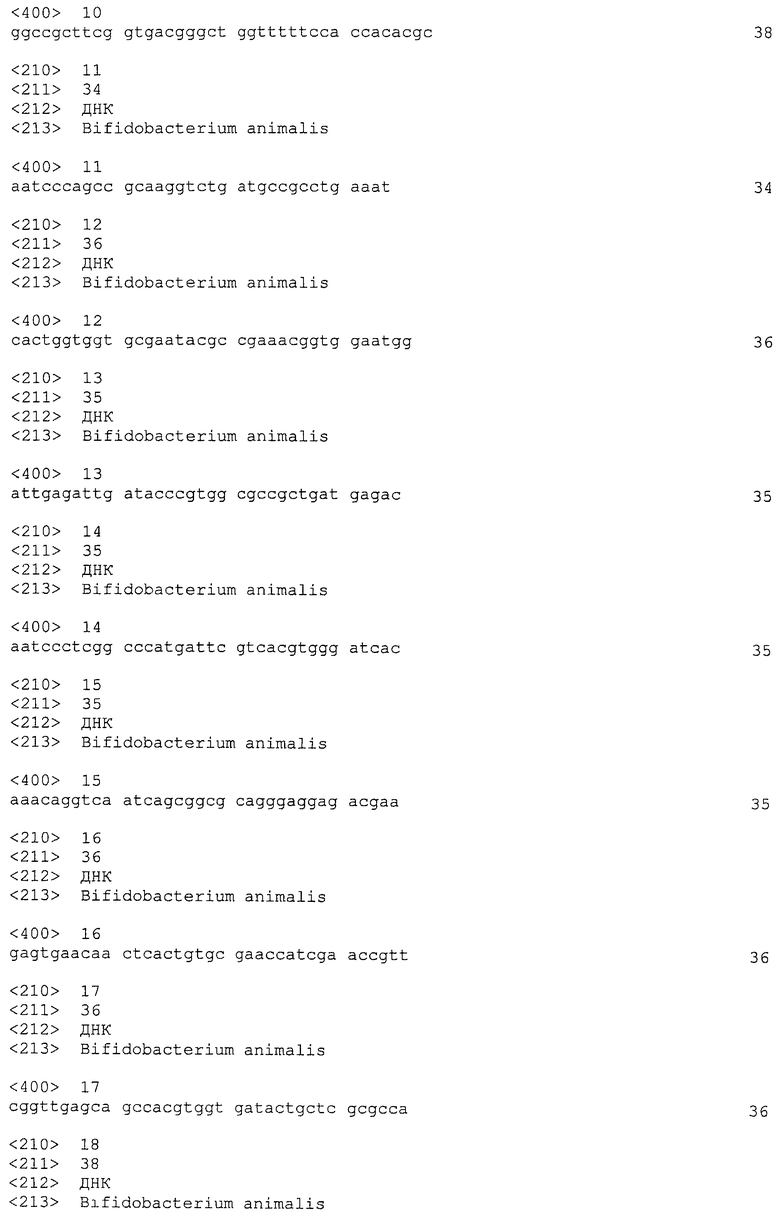




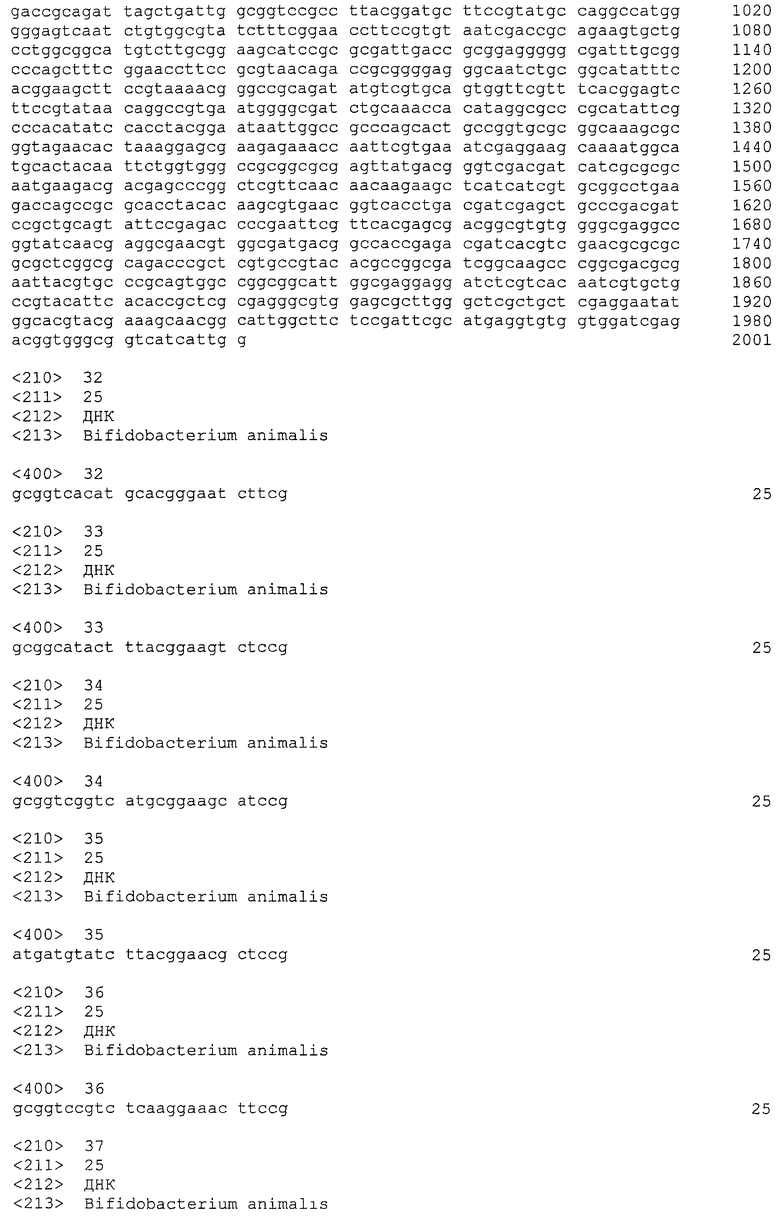
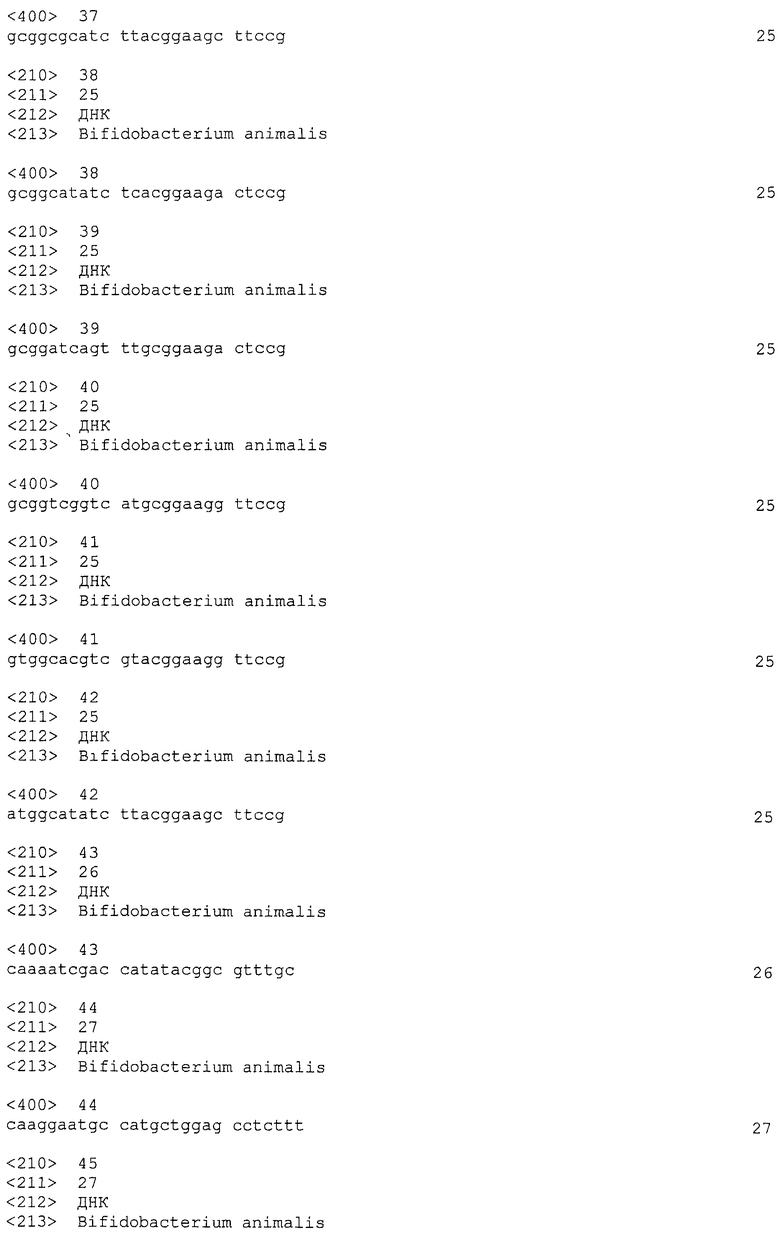
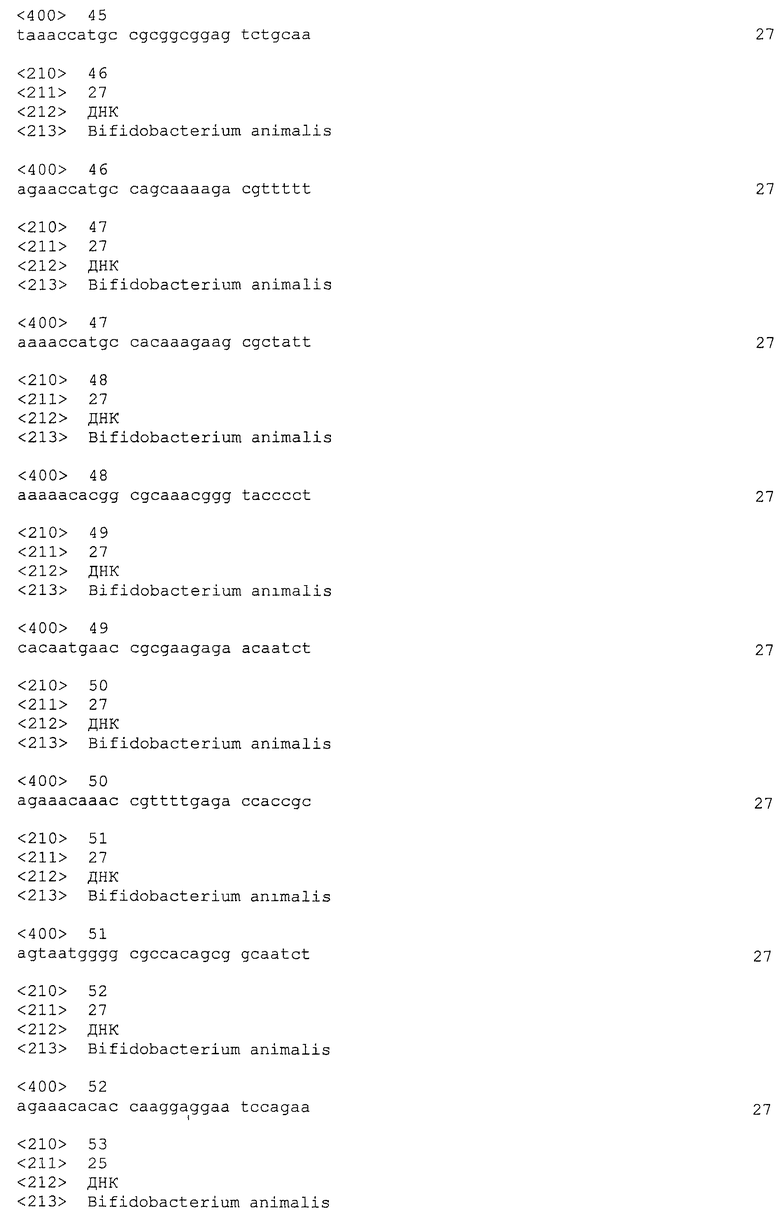
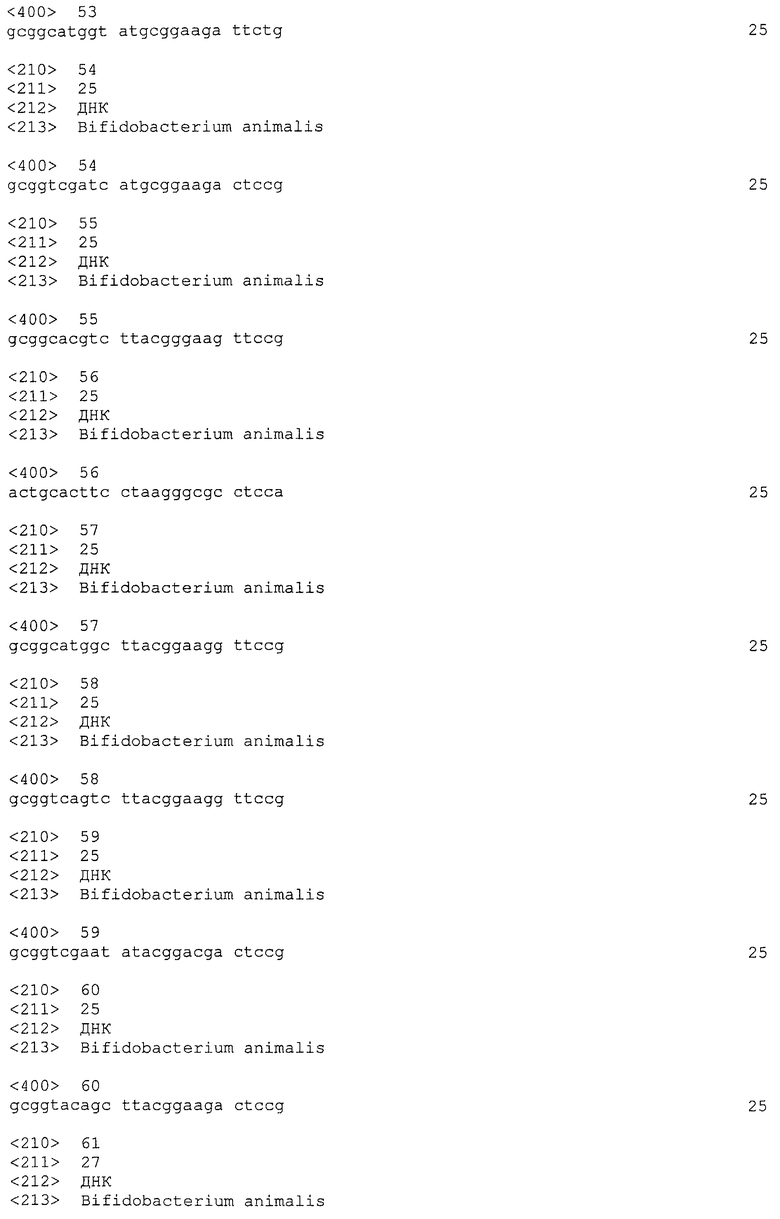
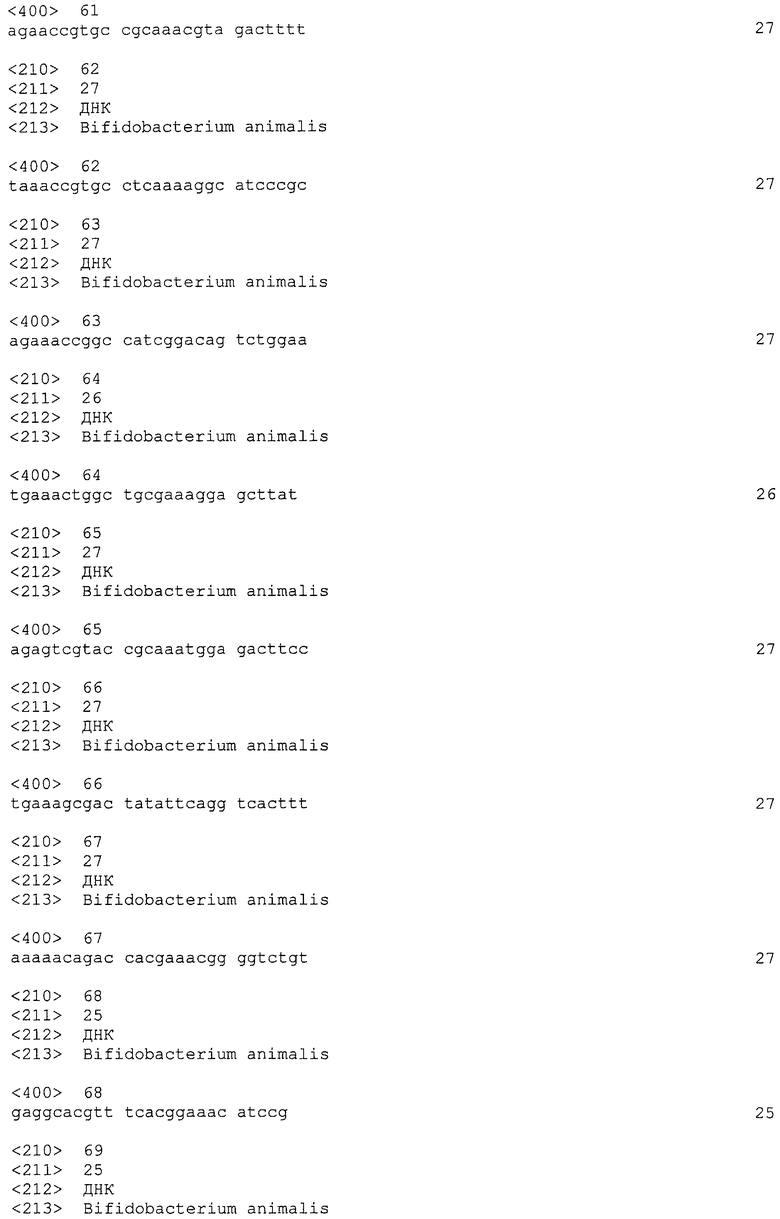
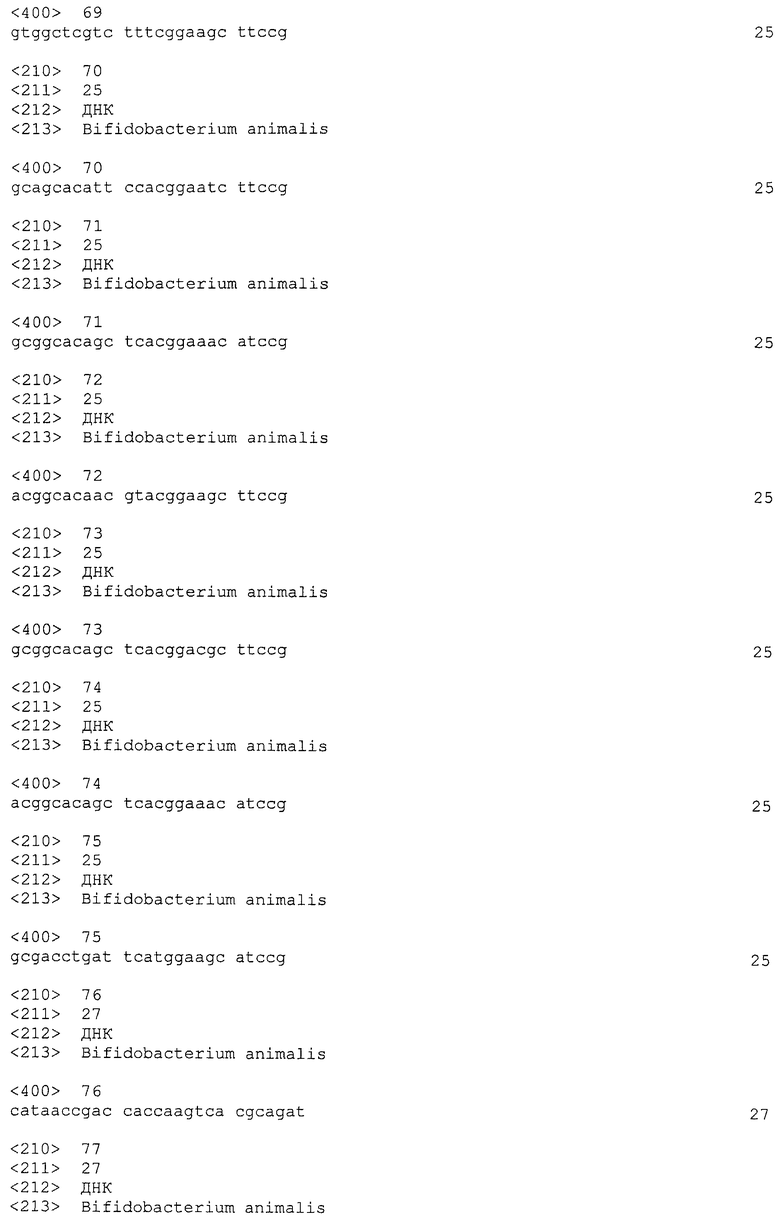
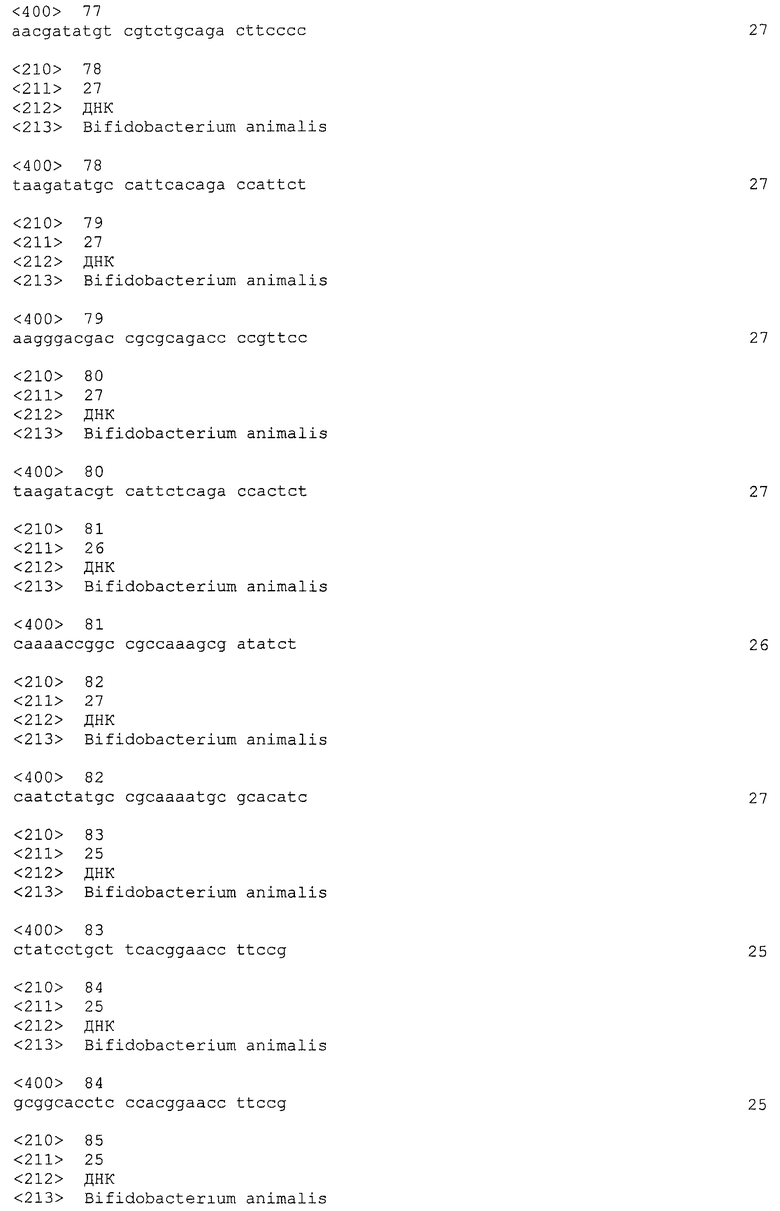
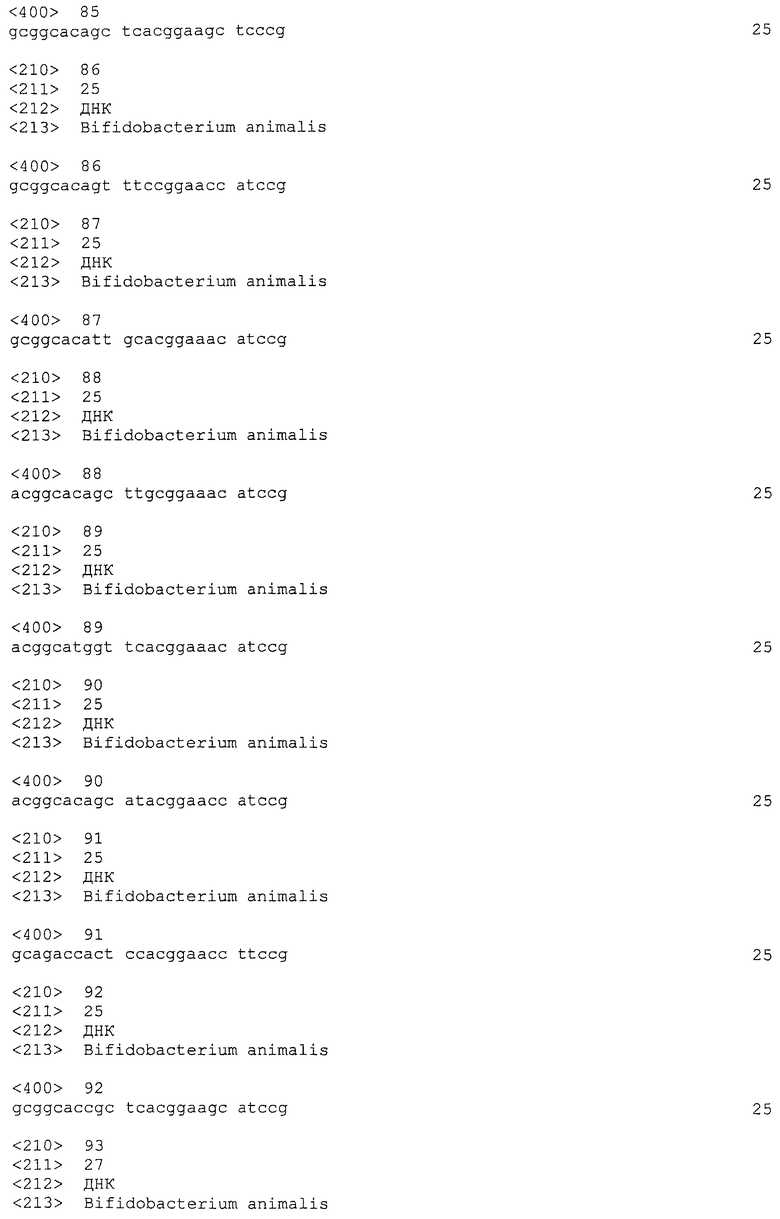
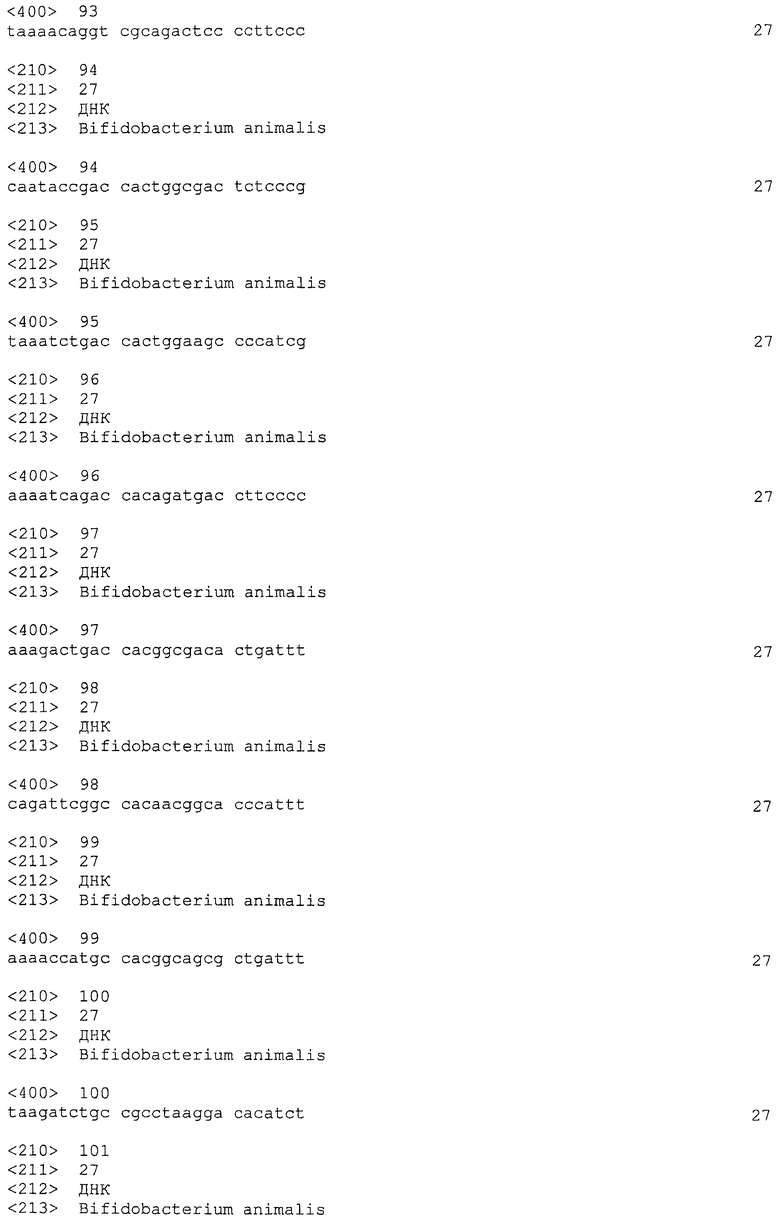

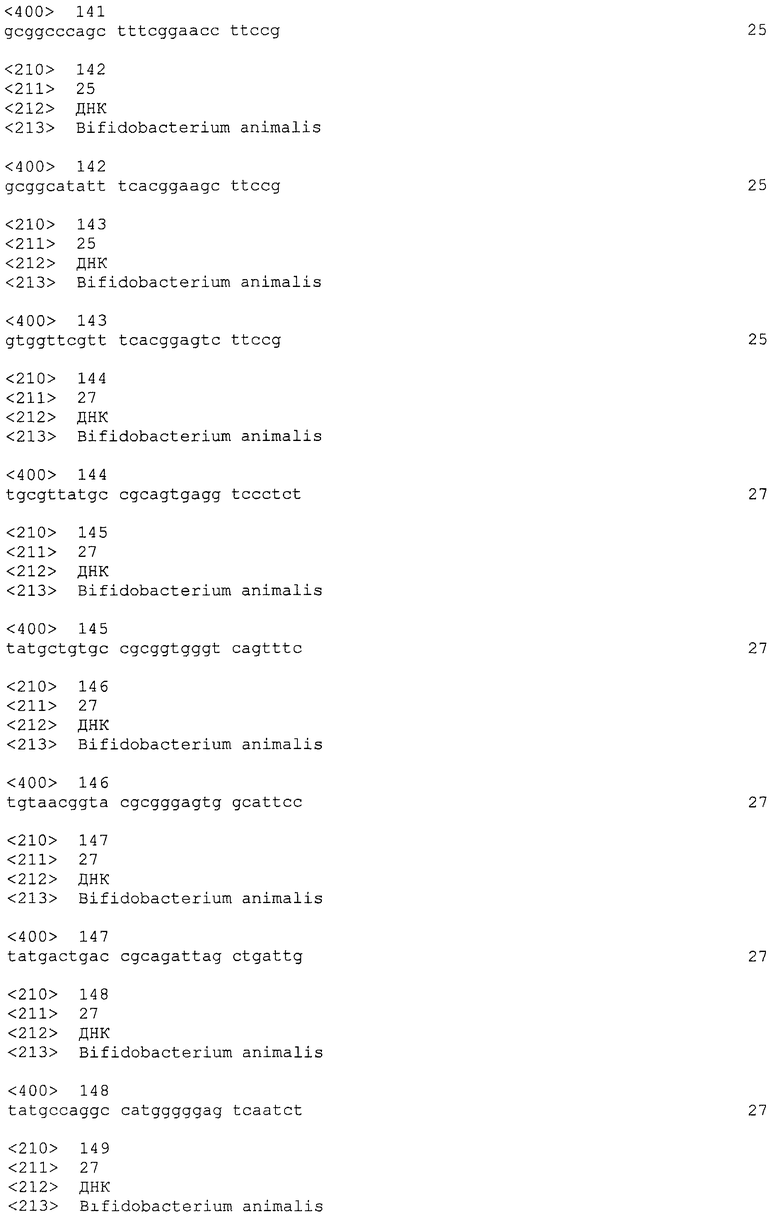










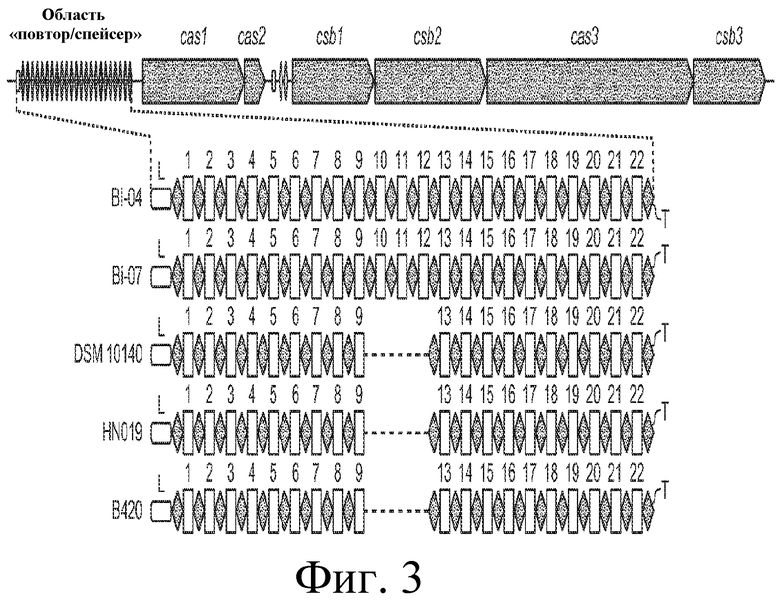

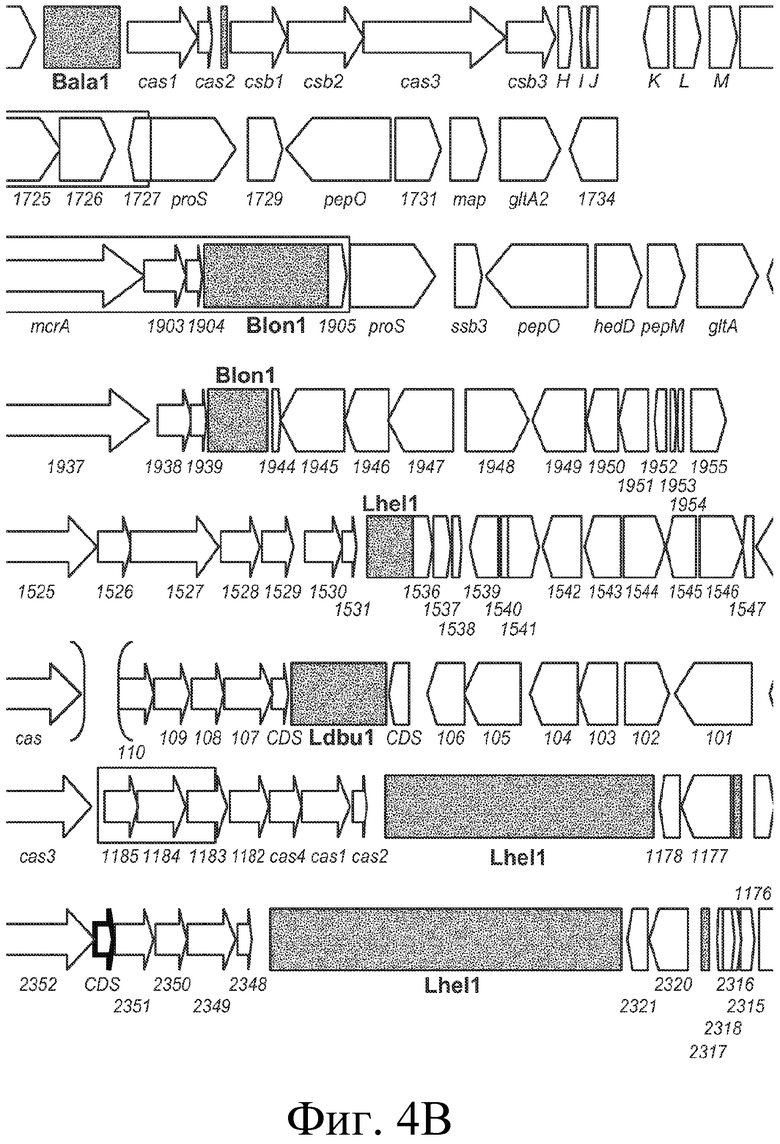
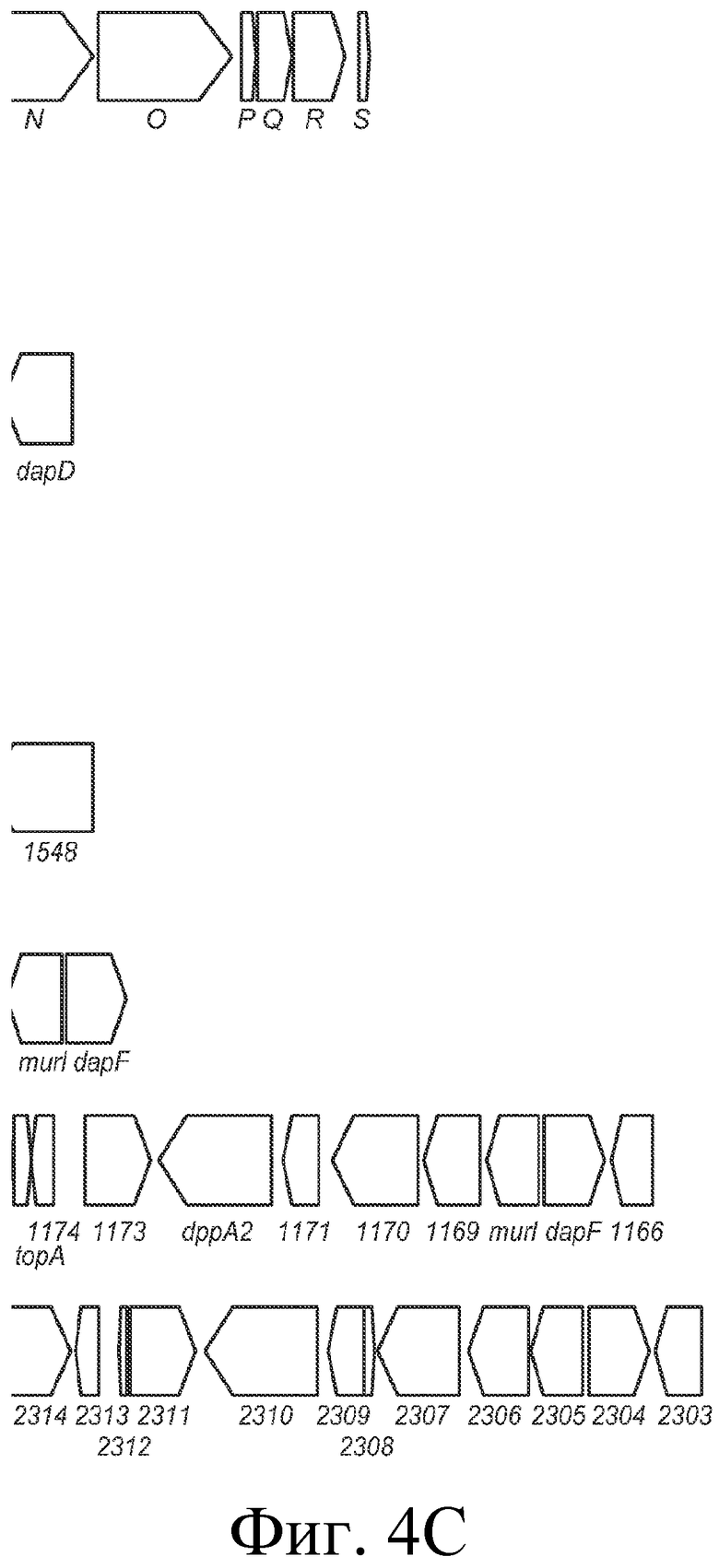







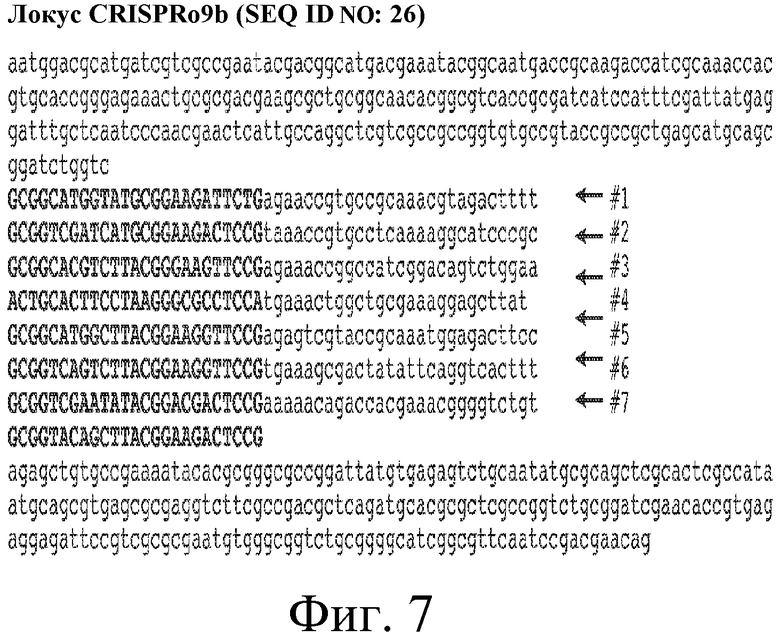



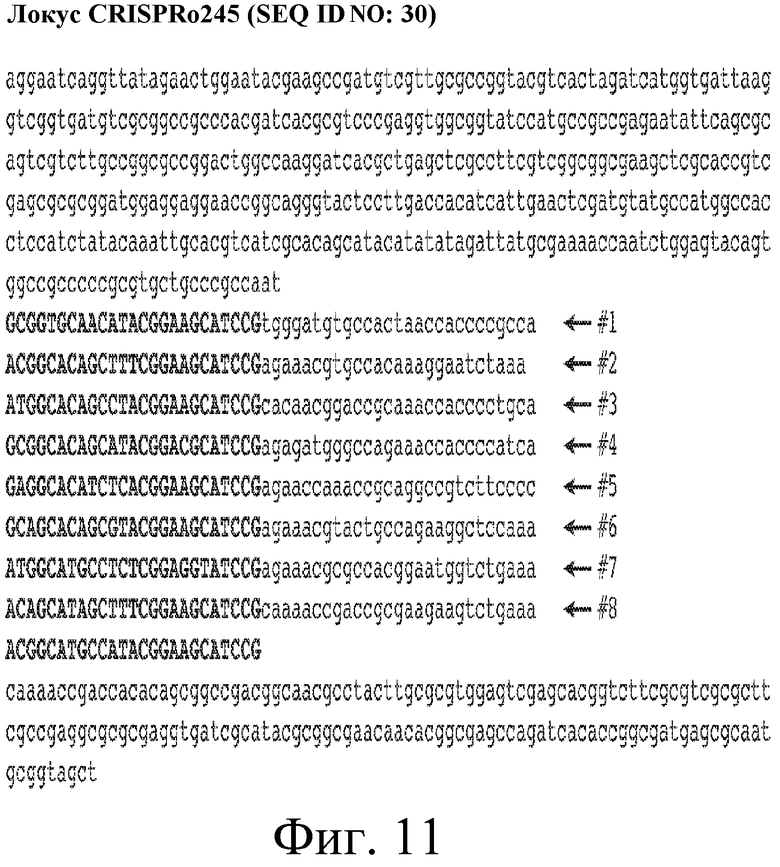

Изобретение относится к биотехнологии и представляет собой выделенную нуклеиновую кислоту для типирования и/или маркировки штаммов Bifidobacterium lactis, состоящую из последовательности локуса CRISPR В.lactis, выбранной из группы, состоящей из a) Balal (SEQ ID NO: 1); b) последовательности повтора Balal, выбранной из последовательности SEQ ID NO: 2 и ее вариантов; c) последовательности спейсера Balal, выбранной из SEQ ID NO:3-24; где варианты последовательности SEQ ID NO: 2 выбраны из группы, состоящей из: замены Т в положении 12, замены Т в положении 14 и замены А в положении 36. Изобретение относится также к вектору, содержащему нуклеиновую кислоту, клетке, трансформированной нуклеиновой кислотой, и к способу типирования штаммов B. lactis. Изобретение позволяет эффективно проводить типирование микроорганизмов. 14 н. и 1 з.п. ф-лы, 12 ил., 3 табл., 1 пр.
1. Выделенная нуклеиновая кислота для типирования и/или маркировки штаммов Bifidobacterium lactis, состоящая из последовательности локуса CRISPR В.lactis, выбранной из группы, состоящей из
a) Bala1 (SEQ ID NO: 1);
b) последовательности повтора Bala1, выбранной из последовательности SEQ ID NO: 2 и ее вариантов;
c) последовательности спейсера Bala1, выбранной из SEQ ID NO:3-24,
где варианты последовательности SEQ ID NO: 2 выбраны из группы, состоящей из: замены Т в положении 12, замены Т в положении 14 и замены А в положении 36.
2. Выделенная нуклеиновая кислота для типирования и/или маркировки штаммов В. lactis, состоящая из последовательности локуса CRISPR В.lactis, выбранной из группы, состоящей из
a) последовательности, способной гибридизоваться в жестких условиях с последовательностью повтора Bala1, выбранной из последовательности SEQ ID NO: 2;
b) последовательности, способной гибридизоваться в жестких условиях с последовательностью спейсера Bala1, выбранной из последовательностей SEQ ID NO: 3-24.
3. Выделенная нуклеиновая кислота по п. 1 или 2, где последовательность выбрана из SEQ ID NO: 13, 14 и 15.
4. Выделенная нуклеиновая кислота для типирования и/или маркировки В.lactis штаммов, состоящая из последовательности повтора Bala1 SEQ ID NO: 2 и последовательности спейсера Bala1, выбранной из SEQ ID NO: 3-24.
5. Вектор для трансформации рекомбинантных штаммов В.lactis, содержащий локус CRISPR, выбранный из группы, состоящей из
a) Bala1 (SEQ ID NO: 1);
b) последовательности повтора Bala1, выбранной из последовательности SEQ ID NO: 2 и ее вариантов;
c) последовательности спейсера Bala1, выбранной из SEQ ID NO: 3-24,
где варианты последовательности SEQ ID NO: 2 выбраны из группы, состоящей из: замены Т в положении 12, замены Т в положении 14 и замены А в положении 36.
6. Вектор для трансформации рекомбинантных штаммов В.lactis, содержащий локус CRISPR, выбранный из группы, состоящей из
a) последовательности, способной гибридизоваться в жестких условиях с последовательностью повтора Bala1, выбранной из последовательности SEQ ID NO: 2;
b) последовательности, способной гибридизоваться в жестких условиях с последовательностью спейсера Bala1, выбранной из последовательностей SEQ ID NO: 3-24.
7. Вектор для трансформации рекомбинантных штаммов В.lactis, содержащий последовательность единицы «повтор-спейсер» Bala1, в которой последовательность повтора выбрана из SEQ ID NO: 2 и ее вариантов, а последовательность спейсера выбрана из SEQ ID NO: 3-24,
где варианты SEQ ID NO: 2 выбраны из группы, состоящей из: замены Т в положении 12, замены Т в положении 14, и замены А в положении 36.
8. Рекомбинантная клетка В.lactis с повышенной устойчивостью к фагу, трансформированная
a) последовательностью повтора Bala1, выбранной из SEQ ID NO: 2 и ее вариантов;
b) последовательностью спейсера Bala1, выбранной из SEQ ID NO: 3-24,
где варианты SEQ ID NO: 2 выбраны из группы, состоящей из: замены Т в положении 12, замены Т в положении 14 и замены А в положении 36.
9. Рекомбинантная клетка В. lactis с повышенной устойчивостью к фагу, трансформированная последовательностью единицы «повтор-спейсер» Bala1, в которой последовательность повтора выбрана из SEQ ID NO: 2 и ее вариантов, а последовательность спейсера выбрана из SEQ ID NO: 3-24,
где варианты последовательности SEQ ID NO: 2 выбраны из группы, состоящей из: замены Т в положении 12, замены Т в положении 14 и замены А в положении 36.
10. Способ получения рекомбинантного штамма В.lactis с повышенной устойчивостью к фагу, включающий
(a) трансформирование бактерий нуклеиновой кислотой, содержащей последовательность спейсера Bala1, выбранную из SEQ ID NO: 3-24;
(b) приведение в контакт трансформированных бактерий с фагом; и
(c) выделение трансформированных бактерий, проявляющих повышенную устойчивость к фагу.
11. Способ типирования штаммов В.lactis, включающий
(a) амплификацию геномной ДНК из представляющего интерес штамма при помощи по меньшей мере одной пары праймеров, где указанная геномная ДНК содержит по меньшей мере часть последовательности локуса CRISPR, где локус CRISPR представляет собой Bala1 (SEQ ID NO: 1), и где каждый праймер из указанной пары является комплементарным по меньшей мере части последовательности локуса CRISPR;
(b) обнаружение ампликона, полученного на этапе (а), при этом указанный обнаруженный ампликон служит признаком конкретного представляющего интерес штамма.
12. Набор для типирования штаммов В.lactis, содержащий
(a) контейнер со смесью для амплификации, содержащей ДНК-полимеразу, буфер амплификации и по меньшей мере одну пару праймеров, где каждый праймер из пары комплементарен части геномной ДНК так, что пара праймеров амплифицирует по меньшей мере часть последовательности повтора или спейсера локуса CRISPR, где локус CRISPR представляет собой Bala1 (SEQ ID NO: 1); и
(b) контейнер со смесью для обнаружения, содержащей зонд, способный гибридизоваться в строгих условиях по меньшей мере с частью локуса CRISPR, амплифицированной с помощью пары праймеров.
13. Способ маркировки штамма В.lactis, включающий:
(a) воздействие фагом на родительский штамм В. lactis;
(b) отбор нечувствительного к фагу мутанта;
(с) сравнение последовательности локуса CRISPR или ее части из родительского штамма и нечувствительного к фагу мутантного штамма;
в результате чего наличие дополнительной единицы «повтор-спейсер» в последовательности локуса CRISPR, нечувствительного к фагу мутанта, указывает на то, что штамм маркирован,
где локус CRISPR представляет собой Bala1 (SEQ ID NO: 1).
14. Выделенная нуклеиновая кислота для трансформации рекомбинантных штаммов В. lactis, состоящая из последовательности локуса CRISPR В.lactis, выбранной из группы, состоящей из
a) Bala1 (SEQ ID NO: 1);
b) последовательности повтора Bala1, выбранной из последовательности SEQ ID NO: 2 и ее вариантов;
c) последовательности спейсера Bala1, выбранной из SEQ ID NO: 3-24,
где варианты последовательности SEQ ID NO: 2 выбраны из группы, состоящей из: замены Т в положении 12, замены Т в положении 14 и замены А в положении 36.
15. Выделенная нуклеиновая кислота для трансформации рекомбинантных штаммов В. lactis, состоящая из последовательности локуса CRISPR В.lactis, выбранной из группы, состоящей из
a) последовательности, способной гибридизоваться в жестких условиях с последовательностью повтора Bala1, выбранной из последовательности SEQ ID NO: 2;
b) последовательности, способной гибридизоваться в жестких условиях с последовательностью спейсера Bala1, выбранной из последовательностей SEQ ID NO: 3-24.
| Пресс для выдавливания из деревянных дисков заготовок для ниточных катушек | 1923 |
|
SU2007A1 |
| GRISSA I., VERGNAUD G., POURCEL C | |||
| The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats | |||
| BMC Bioinformatics vol | |||
| Топка с несколькими решетками для твердого топлива | 1918 |
|
SU8A1 |
| ОДНОВРЕМЕННОЕ ОПРЕДЕЛЕНИЕ, ИДЕНТИФИКАЦИЯ И ДИФФЕРЕНЦИАЦИЯ ЭУБАКТЕРИАЛЬНЫХ ТАКСОНОВ С ПОМОЩЬЮ ГИБРИДИЗАЦИОННОГО АНАЛИЗА | 1995 |
|
RU2154106C2 |
Авторы
Даты
2015-12-10—Публикация
2009-11-06—Подача