Область техники
Настоящее изобретение относится к области биотехнологии, молекулярной биологии и медицины, в частности к белку, обладающему активностью нуклеазы, и различным вариантам применения такого белка. Более конкретно, настоящее изобретение относится к ферменту нуклеазе Cpf1 из бактерии Ruminococcus bromii и молекуле ДНК или РНК, кодирующей указанный фермент. Настоящее изобретение также относится к вектору, содержащему указанную молекулу ДНК, системе CRISPR/Cpf1, содержащую указанную нуклеазу в дополнение к другим элементам CRISPR-кассеты, клетке-хозяину для получения нуклеазы Cpf1, способу получения нуклеазы Cpf1, применению указанной нуклеазы Cpf1 для образования двунитевого или однонитевого разрыва в молекуле ДНК или РНК, способу образования двунитевого или однонитевого разрыва в молекуле ДНК или РНК с использованием указанной нуклеазы, системе доставки, содержащей указанную нуклеазу Cpf1, молекулу ДНК или вектор, комплексу, содержащему указанную нуклеазу Cpf1, молекулу ДНК или вектор, способу модификации целевого локуса ДНК или РНК с использованием указанной нуклеазы, применению указанной нуклеазы Cpf1 для редактирования генома или генной терапии или детекции нуклеиновых кислот.
Уровень техники
Недавние достижения в области технологий секвенирования генома и методов анализа значительно ускорили каталогизацию и картирование генетических факторов, связанных с широким спектром биологических функций и заболеваний. Точные технологии нацеливания на геном необходимы для обеспечения систематической обратной инженерии причинно-следственных генетических вариаций, позволяя избирательно изменять отдельные генетические элементы, а также для продвижения синтетических биологических, биотехнологических и медицинских приложений. Хотя для создания целевых изменений генома доступны такие методы и средства редактирования генома, такие как дизайнерские цинковые пальцы, эффекторы, подобные активаторам транскрипции (TALE) или самонаводящиеся мегануклеазы, остается потребность в новых технологиях и средствах геномной инженерии, которые используют новые стратегии и молекулярные механизмы и являются доступными, простыми в настройке, масштабируемыми и доступными для нацеливания на несколько положений в геноме. Такие решения составят основной ресурс для новых приложений в области геномной инженерии и биотехнологии.
В последние годы было установлено, что редактирование генома, опосредуемое CRISPR-Cas, является полезным инструментом для генной инженерии. Известно, что прокариотические системы CRISPR служат своим хозяевам как адаптивные иммунные системы (Jinek et al., 2012, Science 337: 816-821) и могут быть использованы для быстрой и эффективной генной инженерии (например, Mali et al., 2013, Nat Methods 10: 957-963), требуя только модификации направляющей последовательности.
Системы CRISPR-Cas адаптивного иммунитета бактерий и архей демонстрируют чрезвычайное разнообразие белкового состава и архитектуры геномных локусов. В локусах системы CRISPR-Cas имеется более 50 семейств генов, и не существует строго универсальных генов, что указывает на быструю эволюцию и чрезвычайное разнообразие архитектуры локусов. На сегодняшний день с использованием многостороннего подхода проведена комплексная идентификация гена cas примерно из 395 профилей для 93 белков Cas. Классификация включает профили сигнатурных генов плюс сигнатуры архитектуры локуса. Предложена новая классификация систем CRISPR-Cas, в которой эти системы в целом разделены на два класса: класс 1 с эффекторными комплексами из нескольких субъединиц и класс 2 с эффекторными модулями из одной субъединицы, примером которых является белок Cas9. Новые эффекторные белки, связанные с системами CRISPR-Cas класса 2, могут быть разработаны в качестве мощных инструментов геномной инженерии, и важно предсказывать предполагаемые новые эффекторные белки, а также их конструирование и оптимизацию.
Известна нуклеаза системы CRISPR-Cas9 из бактерии Defluviimonas sp. 20V17, описанная в патенте РФ № 2712492. Известна нуклеаза системы CRISPR-Cas9 из бактерии Pasteurella pneumotropica, описанная в патенте РФ № 2722934. Известны нуклеазы системы CRISPR-Cas9 из бактерий Streptococcus pyogenes, Streptococcus thermophilus, Neisseria meningitidis и Treponema denticola, описанные в патенте РФ № 2704981. Известна нуклеаза системы CRISPR-Cas9 из губок Homoeodictya palmate, описанная в патенте РФ № 2706298. Известно использование нуклеаз системы CRISPR-Cas9 из бактерий Francisella tularensis 1, Francisella tularensis subsp. novicida, Prevotella albensis, Lachnospiraceae bacterium MC2017 1, Butyrivibrio proteoclasticus, Peregrinibacteria bacterium GW2011_GWA2_33_10, Parcubacteria bacterium GW2011_GWC2_44_17, Smithella sp. SCADC, Acidaminococcus sp. BV3L6, Lachnospiraceae bacterium MA2020, Candidatus Methanoplasma termitum, Eubacterium eligens, Moraxella bovoculi 237, Leptospira inadai, Lachnospiraceae bacterium ND2006, Porphyromonas crevioricanis 3, Prevotella disiens и Porphyromonas macacae в искусственной системе, описанной в патенте РФ № 2737537. Известны способы, системы и компоненты для редактирования генома, содержащие полипептид Cpf1 из Acidaminococcus sp. BV3L6, Thiomicrospira sp. XS5, Moraxella bovoculi AAX08_00205, Moraxella bovoculi AAX11_00205 и Lachnospiraceae bacterium MA2020, описанные в международной заявке, опубликованной как WO2017184768.
Изученные в настоящее время нуклеазы систем CRISPR/Cas имеют ограниченное применение в связи с проблемами, связанными со скоростью функционирования, чувствительностью к температурным условиям, и, что наиболее важно - требованиям к последовательности РАМ. Данные ограничения характерны для всех изученных на данный момент Cas нуклеаз. Поиск новых ферментов Cpf1 с другими последовательностями РАМ позволит расширить арсенал имеющихся средств для образования двунитевого или однонитевого разрыва в необходимых, строго определенных местах в молекулах нуклеиновых кислот разных организмов.
Наиболее близкий к заявляемому RbCpf1 ортологу, известный на данный момент, LbCpf1, полученный из Lachnospiraceae bacterium ND2006, аминокислотная последовательность которого имеет 46 % идентичности с аминокислотной последовательностью RbCpf1, в нативном виде распознает канонические 5'-TTTV последовательности PAM (где V - A, G или C), а также неканонические 5'-CTTV, 5'-TCTV, 5'-TTCV последовательности PAM, которые встречаются относительно редко в геномах прокариотических и эукариотических клеток. Такие же PAM распознает и AsCpf1 [Peng Chen, Jin Zhou, Yibin Wan, Huan Liu, Yongzheng Li, Zhaoxin Liu, Hongjian Wang, Jun Lei, Kai Zhao, Yiliang Zhang, Yan Wang, Xinghua Zhang & Lei Yin. (2020). A Cas12a ortholog with stringent PAM recognition followed by low off-target editing rates for genome editing. Genome biology, 21(1), 1-13]. В то же время LbCpf1 не способен распознавать более вырожденные PAM последовательности, которые встречаются гораздо чаще в большинстве геномов с охарактеризованной нуклеотидной последовательностью.
Поэтому в настоящее время сохраняется насущная потребность в разработке новых агентов с улучшенной специфичностью к последовательности-мишени, дающих возможность расщепления мишеней в различных экспериментальных условиях для применения в области генетических исследований, редактирования генома и генной терапии, а также в детекции нуклеиновых кислот.
Технической проблемой, на решение которой направлено заявляемое изобретение, является создание новых инструментов для изменения последовательности геномной ДНК одноклеточных или многоклеточных организмов на основе систем CRISPR-Cas.
Раскрытие изобретения
Техническая проблема решается заявляемой нуклеазой 2 класса V-A подтипа RbCpf1 бактерии Ruminococcus bromii, которая может применяться для внесения направленных изменений в геном как этого, так и других организмов. Направленные изменения могут вноситься путем осуществления разрыва в молекулу ДНК в положении, заданном природным свойством белка, при нуклеотидной последовательности PAM 5'-YYN-3' в указанной молекуле ДНК.
Техническим результатом заявляемой группы изобретений является увеличение пула доступных Cas нуклеаз для геномного редактирования в целом, и расширяющее количество потенциальных мишеней в геноме модифицируемых клеток или другой молекуле ДНК с помощью Cpf1 нуклеаз в частности.
Как известно, в большинстве случаев для эффективного распознавания мишени эффекторным комплексам CRISPR/Cas систем требуется относительно короткая последовательность, примыкающая к протоспейсеру - PAM (англ. Protospacer adjacent motif). PAM-специфичность обусловлена природными свойствами белков - эффекторных нуклеаз в составе комплексов с РНК, и различается у разных представителей Cas нуклеаз. Последнее ограничивает применение каждой отдельно взятой Cas нуклеазы, поскольку количество возможных потенциальных локусов для внесения направленных разрывов в молекулы ДНК обусловлено PAM-специфичностью, выбранной Cas нуклеазы. В целях преодоления этого барьера в рамках данного изобретения была охарактеризована РНК-направляемая ДНК-эндонуклеаза RbCpf1, способная эффективно распознавать PAM-последовательности 5'-YYN-3' в составе молекул ДНК. Особенностями данной нуклеазы RbCpf1 являются: а) наименее строгая из известных Cpf1 PAM-последовательность, исходя из доступных литературных источников; б) расширенный температурный диапазон работы нуклеазы от 20 до 42°C; в) сравнительно небольшой размер среди ортологов нуклеаз Cpf1 (1245 аминокислотных остатков).
Настоящее изобретение относится в целом к новому белку, обладающему активностью нуклеазы, вариантам его применения и содержащим его средствам, в частности, к ферменту нуклеазе Cpf1 из бактерии Ruminococcus bromii. Настоящее изобретение также относится к фрагменту ДНК или РНК, кодирующему указанный фермент, вектору, содержащему указанную молекулу ДНК, системе CRISPR/Cpf1, содержащей указанную нуклеазу и направляющую РНК, клетке-хозяину для получения нуклеазы Cpf1, способу получения нуклеазы Cpf1, применению указанной нуклеазы Cpf1 для образования разрыва в молекуле ДНК или РНК, способу образования разрыва в молекуле ДНК или РНК с использованием указанной нуклеазы, системе доставки, содержащей указанную нуклеазу Cpf1, молекулу ДНК или РНК или вектор, комбинации способов доставки, содержащей указанную нуклеазу Cpf1, молекулу ДНК или РНК или вектор, способу модификации целевого локуса ДНК или РНК с использованием указанной нуклеазы, применению указанной нуклеазы Cpf1 для редактирования генома или генной терапии или детекции нуклеиновых кислот.
Фразу «разрыв вносится в молекулу ДНК в положении, заданном природным свойством белка, при нуклеотидной последовательности PAM в указанной молекуле ДНК» стоит трактовать как внесение разрыва в молекулу ДНК на расстоянии от 0 до 30 нуклеотидов от нуклеотидной последовательности PAM.
Аминокислотная последовательность нуклеазы RbCpf1 представлена в SEQ ID NO: 1.
Последовательность ДНК, кодирующая нуклеазу RbCpf1, представлена в SEQ ID NO: 2.
В частности, в качестве средств для решения задачи настоящего изобретения предложены, обладающий активностью нуклеазы, который содержит аминокислотную последовательность SEQ ID NO: 1; последовательность, которая по меньшей мере на 80% идентична аминокислотной последовательности SEQ ID NO: 1 или фрагменту аминокислотной последовательности SEQ ID NO: 1, и кодирующая такой белок молекула ДНК, содержащая нуклеотидную последовательность, по меньшей мере на 70% идентичную SEQ ID NO: 2.
Их применение позволяет повышать универсальность доступных систем CRISPR-Cas, использовать нуклеазу RbCpf1 для разрезания геномной или плазмидной ДНК в большем количестве специфических сайтов и специфических условий.
В частности, в различных вариантах осуществления настоящее изобретение включает следующие аспекты.
Белок, обладающий активностью нуклеазы, который содержит аминокислотную последовательность SEQ ID NO: 1; последовательность, которая по меньшей мере на 80% идентична аминокислотной последовательности SEQ ID NO: 1; или фрагмент аминокислотной последовательности SEQ ID NO: 1. Предпочтительно когда белок содержит аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности SEQ ID NO: 1, более предпочтительно когда аминокислотная последовательность по меньшей мере на 95% идентична последовательности SEQ ID NO: 1, наиболее предпочтительно когда аминокислотная последовательность по меньшей мере на 98% идентична последовательности SEQ ID NO: 1, очень предпочтительно когда аминокислотная последовательность по меньшей мере на 99% идентична последовательности SEQ ID NO: 1.
Возможен вариант осуществления, согласно которому белок представляет собой природный белок, то есть полученный или используемый непосредственно из бактерии Ruminococcus bromii sp. или внутри клетки Ruminococcus bromii sp.
Возможен вариант осуществления, согласно которому белок представляет собой искусственно синтезированный белок.
Возможен вариант осуществления, согласно которому белок представляет собой рекомбинантный белок, выделенный из гетерологичных систем экспрессии, таких как бактерии, дрожжи и другие эукариотические системы экспрессии.
Возможен вариант осуществления, согласно которому белок получен в бесклеточной системе трансляции.
Возможен вариант осуществления, согласно которому белок представляет собой искусственно гибридный белок.
Молекула ДНК, кодирующая белок, обладающий активностью нуклеазы.
Возможен вариант осуществления, согласно которому молекула ДНК содержит нуклеотидную последовательность, по меньшей мере на 70% идентичную SEQ ID NO: 2, предпочтительно по меньшей мере на 80%, более предпочтительно на 90% идентичную SEQ ID NO: 2. Возможен вариант, когда последовательность идентична по меньшей мере на 95%, предпочтительно на 98%, более предпочтительно, по меньшей мере на 99% идентичную SEQ ID NO: 2. Возможен вариант осуществления, согласно которому молекула ДНК содержит нуклеотидную последовательность SEQ ID NO: 2, предпочтительно молекула ДНК состоит из нуклеотидной последовательности SEQ ID NO: 2. Возможен вариант осуществления, согласно которому молекула РНК состоит из нуклеотидной последовательности, в ходе трансляции которой образуется аминокислотная последовательность SEQ ID NO: 1.
Вектор, содержащий нуклеиновую кислоту согласно заявляемому изобретению изобретения и элементы, обеспечивающие поддержание целостности и/или копийности и/или функциональности вектора. Элементы, обеспечивающие поддержание целостности и/или копийности и/или функциональности молекулы ДНК, включают ориджины репликации, гены устойчивости к антибиотикам, промоторные, операторные, энхансерные и терминаторные регионы генов, регионы связывания транскрипционных факторов. Возможен вариант осуществления, согласно которому вектор является экспрессионным вектором.
Предпочтительно вектор для экспрессии RbCpf1 в клетках человека включает следующие последовательно расположенные элементы: Энхансер цитомегаловируса (CMV) - 235 - 614 п.о.; Промотор цитомегаловируса (CMV) - 615 - 818 п.о.; T7 промотор - 863 - 881 п.о.; Ген RbCpf1 - 917 - 4651 п.о.; Сигнал ядерной локализации нуклеоплазмина NLS - 4652 - 4699 п.о.; Аффинная метка гемаглютинина вируса гриппа HA - 4700 - 4726 п.о.; Сигнал полиаденилирования бычьего гормона роста bGH-poly(A) - 4832 - 5056 п.о.; Ориджин репликации f1 - 5102 - 5530 п.о.; Промотор и энхансер обезьяньего вируса SV40 - 5544 - 5873 п.о.; Ориджин репликации обезьяньего вируса SV40 - 5724 - 5859 п.о.; Ген устойчивости к канамицину, неомицину, G418 NeoR/KanR - 5940 - 6734 п.о.; Сигнал полиаденилирования обезьяньего вируса SV40 - 6908 - 7029 п.о.; Lac оператор - 7102 - 7118 п.о.; Промотор lac - 7126 - 7156 п.о.; Сайт связывания CAP - 7171- 7192 п.о.; Ориджин репликации ColE1/pMB1/pBR322/pUC - 7480 - 8065 п.о.; Ген устойчивости к ампициллину AmpR - 8236 - 9096 п.о.; Промотор гена AmpR - 9097 - 9201 п.о.
Предпочтительно вектор для экспрессии в клетках Escherichia coli включает следующие последовательно расположенные элементы: T7 терминатор - 26-73 п.о.; 6xHis аффинная метка - 140-157 п.о.; Ген RbCpf1 - 159-3898 п.о.; Сайт связывания рибосом из гена 10 фага T7 (RBS) - 3909-3931 п.о.; Lac оператор - 3946-3970 п.о.; T7 промотор - 3971 - 3989 п.о.; lacI промотор - 4302 - 4379 п.о.; Ген lacI - 4380 - 5462 п.о.; Ген rop - 6271 - 6462 п.о.; bom элемент - 6564 - 6706 п.о.; Ориджин репликации ColE1/pMB1/pBR322/pUC - 6892 - 7480 п.о.; Ген устойчивости к канамицину KanR - 7602 - 8417 п.о.; Ориджин репликации фага f1 - 8510 - 8965 п.о. (фиг. 16).
Клетка-хозяин для продуцирования белка, содержащая нуклеиновую кислоту, кодирующую белок, обладающий активностью нуклеазы, или вектор, содержащий нуклеиновую кислоту. Клетка-хозяин представляет собой прокариотическую или эукариотическую клетку. Предпочтительно клетка-хозяин представляет собой клетку бактерии, гриба, растения или животного, в частности, насекомого или млекопитающего.
Клетка-хозяин может быть представлена клеткой, приспособленной для производства и последующего выделения и очистки рекомбинатного белка. Подобные клетки используются в лабораторной практике и известны специалистам. Возможен вариант осуществления, согласно которому клетка-хозяин представляет собой клетку организма, например, из рода Escherichia (Ao, Xiang, Yi Yao, Tian Li, Ting-Ting Yang, Xu Dong, Ze-Tong Zheng, Guo-Qiang Chen, Qiong Wu, and Yingying Guo. "A multiplex genome editing method for Escherichia coli based on CRISPR-Cas12a." Frontiers in microbiology 9 (2018): 2307), Bacillus (Sachla, Ankita J., Alexander J. Alfonso, and John D. Helmann. "A simplified method for CRISPR-Cas9 engineering of Bacillus subtilis." Microbiology spectrum 9, no. 2 (2021): e00754-21), Saccharomyces (Ryan, Owen W., Snigdha Poddar, and Jamie HD Cate. "Crispr-cas9 genome engineering in Saccharomyces cerevisiae cells." Cold Spring Harbor Protocols 2016, no. 6 (2016): pdb-prot086827), Schizosaccharomyces (Ozaki, Aiko, Rie Konishi, Chisako Otomo, Mayumi Kishida, Seiya Takayama, Takuya Matsumoto, Tsutomu Tanaka, and Akihiko Kondo. "Metabolic engineering of Schizosaccharomyces pombe via CRISPR-Cas9 genome editing for lactic acid production from glucose and cellobiose." Metabolic engineering communications 5 (2017): 60-67), Pichia (Liu, Qi, Xiaona Shi, Lili Song, Haifeng Liu, Xiangshan Zhou, Qiyao Wang, Yuanxing Zhang, and Menghao Cai. "CRISPR-Cas9-mediated genomic multiloci integration in Pichia pastoris." Microbial cell factories 18, no. 1 (2019): 1-11), Synechocystis (Cengic, Ivana, Inés C. Cañadas, Nigel P. Minton, and Elton P. Hudson. "Inducible CRISPR/Cas9 allows for multiplexed and rapidly segregated single target genome editing in Synechocystis sp. PCC 6803." bioRxiv (2022)), Chlamydomonas (Shin, Sung-Eun, Jong-Min Lim, Hyun Gi Koh, Eun Kyung Kim, Nam Kyu Kang, Seungjib Jeon, Sohee Kwon et al. "CRISPR/Cas9-induced knockout and knock-in mutations in Chlamydomonas reinhardtii." Scientific reports 6, no. 1 (2016): 1-15), Anabaena (Niu, Tian-Cai, Gui-Ming Lin, Li-Rui Xie, Zi-Qian Wang, Wei-Yue Xing, Ju-Yuan Zhang, and Cheng-Cai Zhang. "Expanding the potential of CRISPR-Cpf1-based genome editing technology in the cyanobacterium Anabaena PCC 7120." ACS synthetic biology 8, no. 1 (2018): 170-180), Arabidopsis (Miki, Daisuke, Wenxin Zhang, Wenjie Zeng, Zhengyan Feng, and Jian-Kang Zhu. "CRISPR/Cas9-mediated gene targeting in Arabidopsis using sequential transformation." Nature Communications 9, no. 1 (2018): 1-9), Solanum (Maioli, Alex, Silvia Gianoglio, Andrea Moglia, Alberto Acquadro, Danila Valentino, Anna Maria Milani, Jaime Prohens et al. "Simultaneous CRISPR/Cas9 Editing of Three PPO Genes Reduces Fruit Flesh Browning in Solanum melongena L." Frontiers in plant science 11 (2020): 607161), Triticum (Tanaka, Jaclyn, Bastian Minkenberg, Snigdha Poddar, Brian Staskawicz, and Myeong-Je Cho. "Improvement of Gene Delivery and Mutation Efficiency in the CRISPR-Cas9 Wheat (Triticum aestivum L.) Genomics System via Biolistics." Genes(Basel) 13, no. 7 (2022): 1180), Nicotiana (Huang, Teng‐Kuei, Brittney Armstrong, Patrick Schindele, and Holger Puchta. "Efficient gene targeting in Nicotiana tabacum using CRISPR/SaCas9 and temperature tolerant LbCas12a." Plant biotechnology journal 19, no. 7 (2021): 1314-1324), Hordeum (Gasparis, Sebastian, Maciej Kała, Mateusz Przyborowski, Leszek A. Łyżnik, Wacław Orczyk, and Anna Nadolska-Orczyk. "A simple and efficient CRISPR/Cas9 platform for induction of single and multiple, heritable mutations in barley (Hordeum vulgare L.)." Plant methods 14, no. 1 (2018): 1-14), Drosophila (Port, Fillip, Maja Starostecka, and Michael Boutros. "Multiplexed conditional genome editing with Cas12a in Drosophila." Proceedings of the National Academy of Sciences 117, no. 37 (2020): 22890-22899), Mus (Pelletier, Stephane, Sebastien Gingras, and Douglas R. Green. "Mouse genome engineering via CRISPR-Cas9 for study of immune function." Immunity 42, no. 1 (2015): 18-27), Rattus (Remy, Séverine, Vanessa Chenouard, Laurent Tesson, Claire Usal, Séverine Ménoret, Lucas Brusselle, Jean-Marie Heslan et al. "Generation of gene-edited rats by delivery of CRISPR/Cas9 protein and donor DNA into intact zygotes using electroporation." Scientific reports 7, no. 1 (2017): 1-13), Homo (Ma, Hong, Nuria Marti-Gutierrez, Sang-Wook Park, Jun Wu, Yeonmi Lee, Keiichiro Suzuki, Amy Koski et al. "Correction of a pathogenic gene mutation in human embryos." Nature 548, no. 7668 (2017): 413-419.; Zhang, Liyang, John A. Zuris, Ramya Viswanathan, Jasmine N. Edelstein, Rolf Turk, Bernice Thommandru, H. Tomas Rube et al. "AsCas12a ultra nuclease facilitates the rapid generation of therapeutic cell medicines." Nature communications 12, no. 1 (2021): 1-15) или других прокариотических или эукариотических клеток, а также представляет собой ткань, орган, органоид или целый организм.
Способ получения белка, обладающего активностью нуклеазы, включает этапы культивирования клетки-хозяина согласно четвертому объекту настоящего изобретения в культуральной среде в условиях, подходящих для продуцирования белка. Например, трансформированные клетки экспрессионного штамма Escherichia coli (такие как BL21(DE3) и его деривативные штаммы (Rosetta, Rosetta2 и аналогичные), либо другие DE3-штаммы) растят в среде Luria-Bertoni (LB) или других подходящих средах (M9, N-C-, LeMaster-Richards (LR)) при 30-37 °С и 100-200 оборотах в минуту до достижения значения оптической плотности 0.4-0.6. Далее к суспензии добавляют ИПТГ (изопропил-β-D-1-тиогалактопиранозид) до концентрации 0.1-1 мМ, снижают температуру до 18-22°С и инкубируют 3-8 часов при 100-150 оборотах в минуту. Осадок клеток, содержащих рекомбинантный белок, суспензируют в стартовом буфере, содержащемдо 100 мМ Tris-HCl pH 7-7.8, до 400 мМ NaCl, 5-10 мМ имидазола, 0-0.2% Triton-X100, 0-5 Мм β-МЭ, 0-1 мМ PMSF при +4 С. Лизат центрифугируют при 20000-28000g в течение 10-60 мин при +4°C и наносят супернатант на металлохелатную колонку Ni-NTA Superflow, предварительно уравновешенную буфером, содержащемдо 100 мМ Tris-HCl pH 7-7.8, до 400 мМ NaCl, 5-10 мМ имидазола, 0-0.1% Triton-X100. Затем колонку промывают последовательно стартовым буфером, промывочным буфером, содержащем до 100 мМ Tris-HCl pH 7-7.8, до 400 мМ NaCl, 40-50 мМ имидазола и элюирующим буфером, содержащем до 100 мМ Tris-HCl pH 7-7.8, до 400 мМ NaCl, 150-500 мМ имидазола. Полученные фракции объединяют, концентрируют и переводят в 20-100 мМ Na-фосфатный буфер с рН 7-7.4 с помощью хроматографии на колонке fast desalting (например, PD-10). Полученный раствор наносят на колонку MonoS 10/100 (GE LifeScience) и проводят элюцию линейным градиентом (0-0.6 М, 10-30 объемов колонки) концентрации NaCl в 20-100-мМ Na-фосфатном буфере рН 7-7.4. Фракции, содержащие целевой белок, объединяют, переводят в буфер для хранения, содержащий 10-50 мМ Tris-HCl pH 7-7.5, 250-500 мМ NaCl, 0-2 мМ ДТТ, 0-10% (об/об) глицерин). Пример хода выделения RbCpf1 проиллюстрирован на фиг. 8-12.
Дополнительно способ может включать этапы выделения и/или очистки продуцированного белка.
Система CRISPR/Cpf1, включающая направляющую (crispr) РНК для белка и содержащая белок, обладающий активностью нуклеазы, либо молекулы ДНК и РНК, кодирующие направляющую РНК и белок.
Применение белка, обладающего активностью нуклеазы, для образования разрыва в молекуле нуклеиновой кислоты, при этом разрыв представляет собой однонитевой разрыв или двунитевой разрыв. Молекула нуклеиновой кислоты представляет собой ДНК или РНК. Предпочтительно разрыв вносится в молекулу ДНК в положении, заданном природным свойством белка, при нуклеотидной последовательности PAM 5'-YYN-3' в указанной молекуле ДНК, или в положении, заданном свойством белка, измененным в ходе внесения направленных или случайных мутаций в аминокислотной SEQ ID NO: 1 или нуклеотидной SEQ ID NO: 2 последовательностях, при нуклеотидной последовательности PAM, распознаваемой белком после внесения направленных или случайных мутаций в SEQ ID NO: 1 и SEQ ID NO: 2.
Способ внесения разрыва в молекулу нуклеиновой кислоты включает использование молекулы нуклеиновой кислоты с белком, обладающим активностью нуклеазы, или молекулы ДНК, кодирующей белок, обладающий активностью нуклеазы. Способ осуществляют in vitro или in vivo. Молекула нуклеиновой кислоты представляет собой молекулу ДНК или молекулу РНК. Предпочтительно разрыв представляет собой однонитевой разрыв или двунитевой разрыв. При этом контакт молекулы нуклеиновой кислоты с белком осуществляют путем введения указанного белка в клетку. Клетка представляет собой эукариотическую или прокариотическую клетку, в частности, клетку бактерии, растения, гриба или животного, например, млекопитающего, в частности, человека.
Способ образования двунитевого разрыва в последовательности геномной ДНК одноклеточного или многоклеточного организма, непосредственно примыкающей к последовательности 5'-YYN-3', или последовательности, содержащей 5'-YYN-3', включающий введение в по меньшей мере одну клетку этого организма эффективного количества: а) белка, обладающего активностью нуклеазы, или молекулы ДНК или РНК, кодирующей белок, обладающий активностью нуклеазы, и б) направляющей РНК, содержащей последовательность, образующую дуплекс с нуклеотидной последовательностью участка геномной ДНК организма, непосредственно примыкающей к нуклеотидной последовательности 5'-YYN-3' или последовательности, содержащей 5'-YYN-3', и взаимодействующей с указанным белком при образовании дуплекса, или последовательности ДНК, кодирующей указанную направляющую РНК.
Под эффективным количеством вводимых в клетку белка и РНК понимается количество белка и РНК, способное осуществлять направленное, с помощью указанной РНК, внесение двунитевого разрыва в последовательность ДНК, непосредственно примыкающей к последовательности PAM на ДНК, с помощью указанного белка.
Система доставки, содержащая белок, молекулу ДНК или вектор. При этом вектор представляет собой молекулу ДНК, содержащую вместе с элементами, кодирующими белок Cpf1 и/или направляющую РНК, элементы, необходимые для поддержания целостности и/или копийности и/или функциональности данной молекулы ДНК. Например, в случае плазмидного вектора элементами, необходимыми для поддержания целостности и/или копийности и/или функциональности плазмидного вектора, являются ориджин репликации, ген или гены устойчивости к антибиотикам, а также регуляторные элементы для генов в составе плазмидного вектора, например, промоторы, операторы, энхансеры и терминаторы транскрипции, либо комбинации этих элементов.
Возможен вариант комбинации методов доставки к целевой ДНК нуклеазы Cpf1, молекулы ДНК или РНК, кодирующей нуклеазу Cpf1, молекулы направляющей РНК или ДНК, кодирующей направляющую РНК, а также вектора, кодирующего нуклеазу Cpf1 и/или направляющую РНК. При этом нуклеаза Cpf1 доставляется в клетку в виде гена в составе молекулы ДНК, например, вектора, а направляющая или направляющие РНК доставляются в форме РНК.
Возможен вариант осуществления, согласно которому нуклеаза Cpf1 доставляется в клетку в виде белка или РНК, кодирующей нуклеазу Cpf1, а направляющая или направляющие РНК доставляются в виде гена в составе молекулы ДНК, в том числе вектора.
Возможен вариант осуществления, согласно которому нуклеаза Cpf1 доставляется в клетку в виде белка или РНК, кодирующей нуклеазу Cpf1, а направляющая или направляющие РНК доставляются в форме РНК.
Возможен вариант осуществления, согласно которому нуклеаза Cpf1 доставляется в клетку в виде гена в составе молекулы ДНК, например, вектора, и направляющая или направляющие РНК доставляются в виде гена или генов в составе молекулы ДНК, в том числе, вектора.
Способ модификации целевого локуса нуклеиновой кислоты, включающий доставку к указанному локусу и использование для внесения изменений в ДНК белка Cpf1, молекулы ДНК, РНК или вектора и подходящей направляющей РНК или системы CRISPR/Cpf1.
Применение в области биотехнологии, молекулярной биологии и медицины белка, молекулы ДНК или вектора заявляемого изобретения для редактирования генома. Предпочтительно редактируют геном одноклеточного или многоклеточного организма. При этом организм представляет собой прокариотический или эукариотический организм, в частности, относится к простейшим, бактериям, растениям, грибам, животным, в частности, представляет собой млекопитающее, такое как человек.
Возможен вариант осуществления, согласно которому применение осуществляют для коррекции генетического дефекта в виде геномных мутаций в организме, включающих в себя однонуклеотидные полиморфизмы и/или инсерции и/или делеции и/или транслокации и/или инверсии, в частности, в терапевтических целях, путем внесения однонитевых или двунитевых разрывов в молекулы ДНК в присутствии или без такового донорной последовательности, или в виде нарушений экспрессии генов, таких как снижение или повышение экспрессии генов, путем направленного изменения экспрессии, опосредованного использованием белка Cpf1, молекулы ДНК, РНК или вектора и подходящей направляющей РНК или системы CRISPR/Cpf1.
Белок, молекула ДНК или РНК или вектор заявляемого изобретения для применения для редактирования генома одноклеточного или многоклеточного организма. При этом организм представляет собой прокариотический или эукариотический организм, в частности, относится к простейшим, бактериям, растениям, грибам, животным, в частности, представляет собой млекопитающее, такое как человек. Предпочтительно применение осуществляют для коррекции генетического дефекта в виде геномных мутаций в организме, включающих в себя однонуклеотидные полиморфизмы и/или инсерции и/или делеции и/или транслокации и/или инверсии, в частности, в терапевтических целях, путем внесения однонитевых или двунитевых разрывов в молекулы ДНК в присутствии или без такового донорной последовательности, или в виде нарушений экспрессии генов, таких как снижение или повышение экспрессии генов, путем направленного изменения экспрессии, опосредованного использованием белка Cpf1, молекулы ДНК, РНК или вектора заявляемого изобретения и подходящей направляющей РНК или системы CRISPR/Cpf1.
Способ редактирования генома одноклеточного или многоклеточного организма, включающий внесение в, по меньшей мере, одну клетку указанного организма белка, молекулы ДНК, вектора или системы CRISPR/Cpf1 заявляемого изобретения для внесения разрыва в геномную ДНК указанного организма.
Возможен вариант осуществления, дополнительно включающий встраивание в геном экзогенной последовательности ДНК за счет гомологичной репарации, путем использования донора гомологии в виде молекулы ДНК.
Возможен вариант осуществления, согласно которому экзогенная последовательность ДНК представляет собой один или более участков двунитевой или однонитевой ДНК из генома организма, отличного от организма, геном которого редактируют, или из генома организма, геном которого редактируют, измененный по одному или большему числу положений нуклеотидов, или искусственно синтезированный участок или участки ДНК.
Возможен вариант осуществления, согласно которому организм представляет собой прокариотический или эукариотический организм, в частности, относится к простейшим, бактериям, растениям, грибам, животным, в частности, представляет собой млекопитающее, такое как человек.
Возможен вариант осуществления, согласно которому редактирование осуществляют в терапевтических целях. Например, с целью изменения уровня экспрессии генов, в том числе полного выключения экспрессии путем внесения мутаций в кодирующую или регуляторную области этих генов.
Белок, молекула ДНК, РНК или вектор или система CRISPR/Cpf1 согласно заявляемого изобретения для применения в терапии.
Комбинация способов доставки белка для образования разрывов в молекуле нуклеиновой кислоты, содержащая белок или молекулу ДНК или РНК, кодирующих нуклеазу, согласно заявляемого изобретения.
Белок, молекула ДНК или РНК или система CRISPR/Cpf1, заявляемого изобретения для применения в целях детекции нуклеиновой кислоты.
Возможен вариант осуществления, согласно которому система CRISPR/Cpf1 вносит разрыв в молекулу целевой нуклеиновой кислоты в присутствии молекулы ДНК или РНК, меченной детектируемой меткой или метками. Например, флуоресцентными (Broughton J.P., Deng X., Yu G., Fasching C.L., Servellita V., Singh J., Miao X., Streithorst J.A., Granados A., Sotomayor-Gonzalez A., Zorn K., Gopez A., Hsu E., Gu W., Miller S., Pan C.Y., Guevara H., Wadford D.A., Chen J.S., Chiu C.Y. CRISPR-Cas12-based detection of SARS-CoV-2 // Nat.Biotechnol. - 2020. - V. 38. - № 7. - P. 870-874), такими как FAM, HEX, ROX, VIC, Cy5, Cy3 и другими, люменесцентными (Li, Cheng-Yu, Bei Zheng, Jiang-Tao Li, Jia-ling Gao, Yu-Heng Liu, Dai-Wen Pang, and Hong-Wu Tang. "Holographic optical tweezers and boosting upconversion luminescent resonance energy transfer combined clustered regularly interspaced short palindromic repeats (CRISPR)/Cas12a biosensors." ACS nano 15, no. 5 (2021): 8142-8154), например, апконверсионными наночастицами, колориметрическими, такими как наночастицы золота, (Jiang Y., Hu M., Liu A.A., Lin Y., Liu L., Yu B., Zhou X., Pang D.W. Detection of SARS-CoV-2 by CRISPR/Cas12a-Enhanced Colorimetry // ACS Sensors. - 2021. - V. 6. - № 3. - P. 1086-1093) молекулами в качестве детектируемых меток.
Краткое описание чертежей
Изобретение поясняется следующими чертежами.
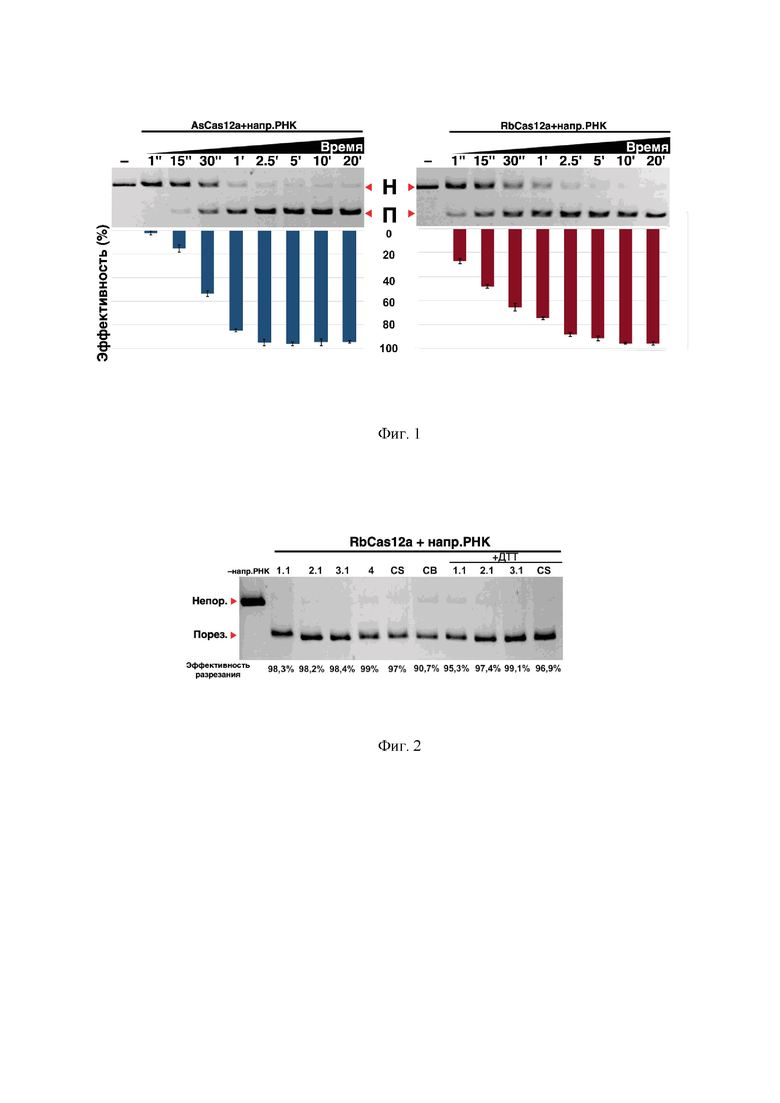
На фиг. 1 представлена электрофореграмма in vitro разрезания для определения времени разрезания ДНК-матриц нуклеазой RbCpf1 по сравнению с AsCpf1. Эффективность разрезания ДНК-мишени в зависимости от времени инкубации с помощью RbCpf1 и AsCpf1. В реакции добавляли эквимолярные количества рекомбинантных AsCpf1 или RbCpf1. Соотношение ДНК-матрица:Cpf1:РНК 1:3:30. Концентрация crРНК 0.5 мМ. Показаны средние эффективности разрезания и стандартные отклонения, рассчитанные по трем независимым экспериментам. Н. - негидролизованные фрагменты ДНК-матрицы, П. - гидролизованные фргаменты ДНК-матрицы, напр.РНК -направляющая РНК. Дорожки с реакциями без напр.РНК обозначены минусом «-».
На фиг. 2 представлена электрофореграмма in vitro разрезания нуклеазой RbCpf1 для определения активности RbCpf1 в различных буферах. Эффективности разрезания RbCpf1 в различных буферах. Реакции проводили в присутствии RbCpf1 и 0.5 мМ направляющей РНК. Соотношение ДНК-матрицы: RbCpf1: РНК 1:3:30. Исследовали активности в NEBuffer 1.1 (1.1), NEBuffer 2.1 (2.1), NEBuffer 3.1 (3.1), NEBuffer 4 (4), CutSmart (CS) (New England Biolabs) и буфера для разрезания (CB) с добавлением или без добавления 5 мМ дитиотрейтола (ДТТ). Снизу показаны средние значения эффективности разрезания по результатам трех независимых экспериментов. Непор. - непорезанные фрагменты ДНК, порез. - порезанные фрагменты ДНК, напр.РНК -направляющая РНК.
На фиг. 3 представлена электрофореграмма in vitro разрезания нуклеазой RbCpf1 для определения температурного оптимума работы нуклеазы. Влияние температуры на эффективность разрезания in vitro RbCpf1. Разрезание ДНК-мишени с помощью RbCpf1, в присутствии 0.5 мМ направляющей РНК при соотношении ДНК-матрицы:RbCpf1:РНК 1:3:30. Показаны средние эффективности разрезания и стандартные отклонения, рассчитанные по трем независимым экспериментам. Непор. - непорезанные фрагменты ДНК, порез. - порезанные фрагменты ДНК, напр.РНК -направляющая РНК.
На фиг. 4 представлены результаты определения PAM последовательности нуклеазы RbCpf1. График полученной при анализе данных высокопроизводительного секвенирования (NGS) консенсусной PAM-последовательности RbCpf1 - 5′-YYN-3′. Нумерация нуклеотидов по оси абсцисс соответствует 5′-3′ направлению, начиная с первого вырожденного нуклеотида по нетаргетной цепи PAM-библиотеки (он же нуклеотид -8). Напр.РНК - направляющая РНК. Как видно из графика, для RbCpf1 последовательность PAM была определена как трехнуклеотидный мотив 5′-YYN-3′, где Y-пиримидиновый нуклеотид (либо С, либо Т), а N - любой нуклеотид.
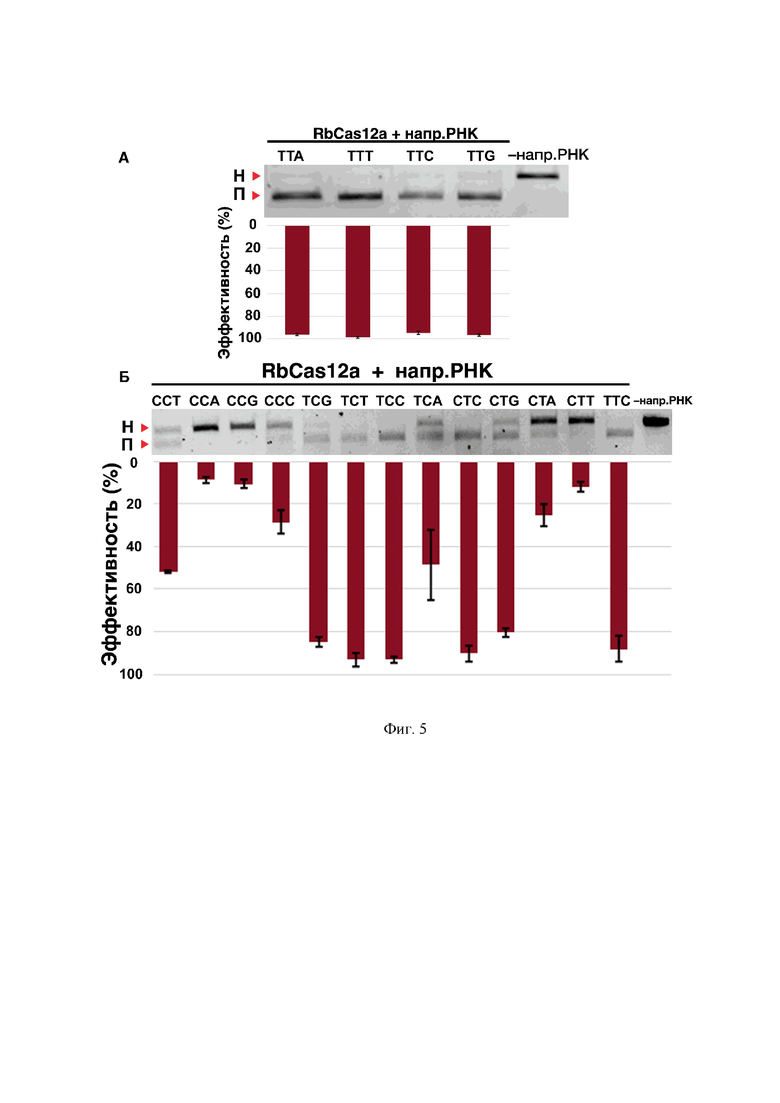
На фиг. 5 представлена электрофореграмма in vitro разрезания для подтверждения PAM-специфичности нуклеазы RbCpf1. Изучение PAM-специфичности нуклеазы RbCpf1 в условиях in vitro. На верхней части (А) показана электрофореграмма разрезаний ДНК-матрицы с одним и тем же протоспейсером, но разными нуклеотидами в последней 3′-концевой позиции PAM (позиция -1). В качестве контроля использовалась матрица с PAM 5′-TTG без добавления направляющей РНК (-напр.РНК). На нижней части (Б) показана электрофореграмма разрезаний pUC119 с использованием различных направляющих РНК на протоспейсеры с различными поcледовательностями PAM вида 5′-YYN-3′, на последней дорожке контрольная матрица без добавления направляющей РНК. Под каждой дорожкой на обоих частях рисунка столбики, иллюстрирующие средние значения относительных эффективностей разрезания в % по трем повторам ± стандартное отклонение. Н. - непорезанные фрагменты ДНК, П. - порезанные фрагменты ДНК, напр.РНК -направляющая РНК.
На фиг. 6 представлены результаты испытания нуклеазы RbCpf1 в клетках человека с формированием делеции в гене DNMT1. Редактирование гена DNMT1 с помощью двух направляющих РНК (crРНК1 и crРНК2) и RbCpf1. Сверху: электрофореграмма ампликонов фрагмента гена DNMT1 после трансфекции культуры клеток HEK293T тремя плазмидными векторами, кодирующими соответственно: crРНК1, crРНК2 и RbCpf1. Три независимых эксперимента пронумерованы 1-3 на второй и четвертый день после трансфекции. Показана схема ПЦР-продукта, содержащего два протоспейсера (№1 и №2), узнаваемых соответственно направляющими РНК crРНК1 и crРНК2. Снизу: детальная карта делеции с нуклеотидной последовательностью, выявленной с помощью секвенирования по Сэнгеру. Сайты посадки crРНК выделены черным цветом, PAM-последовательности выделены красным. Красно-белыми стрелками показаны выравненные хроматограммы после секвенирования по Сэнгеру; красные части стрелок - выравнивающиеся последовательности, белые - отсутствующие в хроматограммах. Продемонстрирована активность RbCpf1 в клетках человека с формированием делеции в гене DNMT1.
На фиг. 7 представлено выравнивание аминокислотных последовательностей нуклеаз CeCpf1 (CeCas12a), LbCpf1 (LbCas12a), RbCpf1 (RbCas12a).
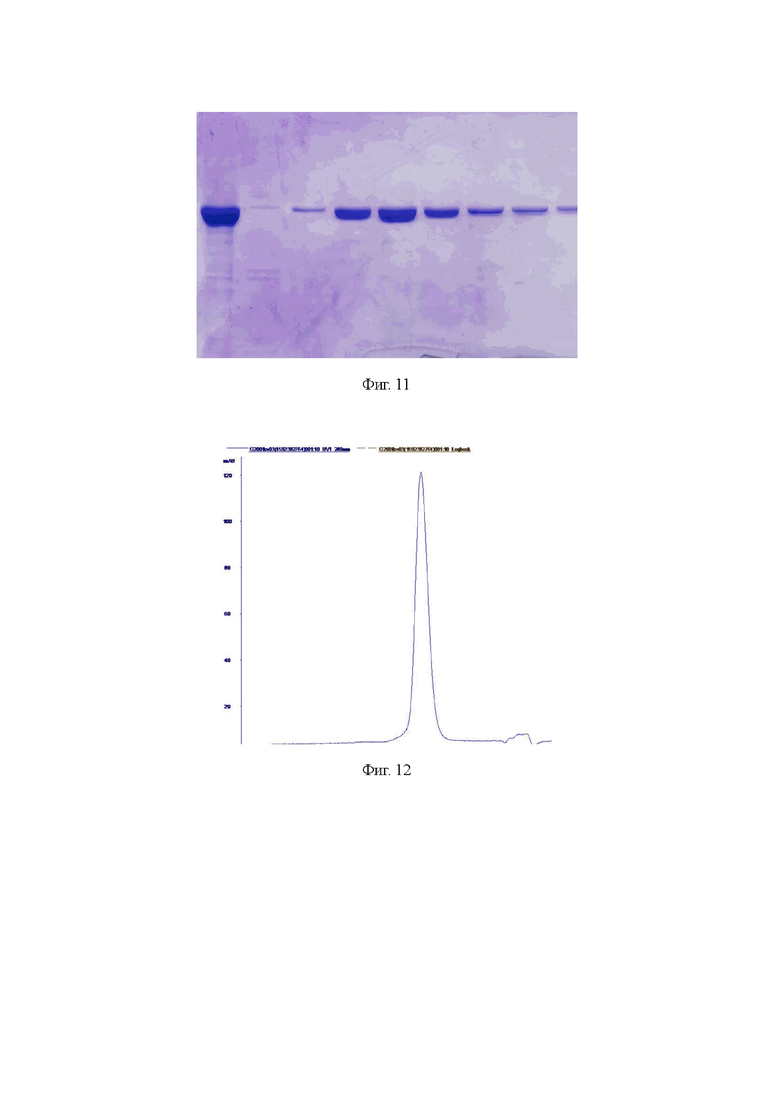
На фиг. 8-12 представлена очистка RbCpf1 хроматографическим методом. На фиг. 8 представлена электрофореграмма в денатурирующем полиакриламидном геле фракций, полученных после металло-аффинной хроматографии. Дорожки слева направо: 1 - клеточный лизат, 2 - осадок, 3 - проскок через колонку, 4 - фракция W с колонки, 5 - фракция Е с колонки, 6 - маркер молекулярных масс PageRuler Plus (ThermoFisher Scientific). На фиг. 9 представлено распределение целевого белка RbCpf1 в промывочной W и элюирующей E фракциях. Левый пик - проскок. По оси абсцисс показано время в минутах, по оси ординат - оптическая плотность в mAU. На фиг. 10 представлено распределение элюата с целевым белком RbCpf1. По оси абсцисс указан объем в мл, по оси ординат - оптическая плотность в mAU. На фиг. 11 представлена электрофореграмма в денатурирующем полиакриламидном геле фракций, полученных после катионно-обменной хроматографии. Дорожки слева направо: 1 - нанесение на колонку, 2-9 - фракции, 10 - маркер молекулярных масс PageRuler Plus (ThermoFisher Scientific). На фиг. 12 представлено распределение целевого белка RbCpf1 в ходе гель-фильтрации. По оси абсцисс указан объем в мл, по оси ординат - оптическая плотность в mAU.
На фиг. 13 представлена электрофореграмма in vitro разрезания ДНК-матрицы нуклеазами RbCpf1, RbCpf1_mut, RbCpf1_mut2. Дорожки слева направо: негативный контроль (без нуклеазы), разрезание с RbCpf1, разрезание с RbCpf1_mut, разрезание с RbCpf1_mut2, маркер молекулярных масс ThermoFisher UltraLow Range. Нижние полосы - направляющая РНК.
На фиг. 14 выравнивание аминокислотных последовательностей RbCpf1 и RbCpf1_mut. Гомология двух последовательностей 97%.
На фиг. 15 выравнивание аминокислотных последовательностей RbCpf1 и RbCpf1_mut2. Гомология двух последовательностей 97%.
На фиг. 16 представлена карта примера плазмидного экспрессионного вектора для экспрессии в клетках Escherichia coli нуклеазы RbCpf1 с целью дальнейшего выделения и очистки белка.
На фиг. 17 представлена карта примера плазмидного вектора для экспрессии RbCpf1 в клетках человека.
Осуществление изобретения
Ниже представлено более подробное описание заявляемого изобретения. Настоящее изобретение может подвергаться различным изменениям и модификациям, понятным специалисту на основе прочтения данного описания. Такие изменения не ограничивают объем притязаний. Например, могут быть изменены неконсервативные аминокислотные остатки, изменения в которых не оказывают влияния на функцию белка.
Как указано выше, задача настоящего изобретения решена при помощи белка, обладающего активностью нуклеазы, содержащего аминокислотную последовательность SEQ ID NO: 1, или содержащего аминокислотную последовательность, которая по меньшей мере на 80% идентична аминокислотной последовательности SEQ ID NO: 1 и имеет отличия по сравнению с SEQ ID NO: 1 только в аминокислотных остатках, изменения в которых не приводят к изменению или уничтожению функции белка. Неконсервативные аминокислотные остатки RbCpf1 представлены на фиг. 7. Изменения могут быть представлены, например, заменами неконсервативных аминокислотных остатков на гомологичные. Замены аминокислотных остатков на гомологичные можно провести методами, известными специалистам в области генной инженерии, например с помощью сайт-направленного мутагенеза методом ПЦР последовательностей ДНК (Reikofski, Julia, and Bernard Y. Tao. "Polymerase chain reaction (PCR) techniques for site-directed mutagenesis." Biotechnology advances 10, no. 4 (1992): 535-547), кодирующих белок нуклеазу RbCpf1. Этот белок может образовывать разрыв в молекуле ДНК в точке, непосредственно примыкающей к последовательности 5'-YYN-3', или последовательности, содержащей 5'-YYN-3' в указанной молекуле ДНК. В некоторых вариантах осуществления образование двунитевого разрыва in vitro в молекуле ДНК происходит за 1 секунду. В предпочтительных вариантах изобретения белок применяется в качестве средства для образования двунитевого разрыва in vitro на протяжении 20 минут.
Дополнительно предложен способ образования двунитевого разрыва в последовательности геномной ДНК одноклеточного или многоклеточного организма, примыкающей к последовательности 5'-YYN-3', включающего введение в по меньшей мере одну клетку этого организма эффективного количества: а) белка, содержащего аминокислотную последовательность SEQ ID NO: 1, или нуклеиновой кислоты, кодирующей белок, содержащий аминокислотную последовательность SEQ ID NO: 2, и б) направляющей РНК, содержащей последовательность, образующую дуплекс с нуклеотидной последовательностью участка геномной ДНК организма, примыкающей к нуклеотидной последовательности 5'-YYN-3' или содержащей последовательность 5'-YYN-3', и взаимодействующей с указанным белком при образовании дуплекса, или последовательности ДНК, кодирующей указанную направляющую РНК; при этом взаимодействие указанного белка с направляющей РНК и нуклеотидной последовательностью, содержащей 5'-YYN-3', приводит к образованию двунитевого разрыва в последовательности геномной ДНК. В некоторых вариантах изобретения данный способ характеризуется тем, что дополнительно включает введение экзогенной последовательности ДНК одновременно с направляющей РНК.
Изобретение может быть использовано как для разрезания целевой ДНК in vitro, так и для модификации генома живого организма. Модификация генома может проводиться путем разрезания генома в соответствующем сайте и вставки экзогенной последовательности ДНК за счет гомологичной репарации.
Введение в клетки нуклеиновых кислот, кодирующих RbCpf1 и/или направляющие РНК и/или сами направляющие РНК, может быть осуществлено путем трансфекции или трансформации клеток различными методами, известными специалистам (липофекция, электропорация, вирусная трансдукция, микроинъекция и т.д.).
Введение в клетки белка RbCpf1 может быть осуществлено путем трансфекции или трансформации клеток различными методами, известными специалистам (липофекция, электропорация, микроинъекция и т.д.).
Введение в клетки комплексов RbCpf1 с направляющими РНК может быть осуществлено путем трансфекции или трансформации клеток различными методами, известными специалистам (липофекция, электропорация, микроинъекция и т.д.).
В качестве экзогенной последовательности ДНК может быть использован любой участок двунитевой или однонитевой ДНК из генома организма, отличного от организма, используемого при введении (или смесь таких участков между собой и с другими фрагментами нуклеиновых кислот), при этом этот участок (или смесь участков) предназначен для интеграции в место разрыва в целевой ДНК, образованного под действием нуклеазы RbCpf1. В некоторых вариантах изобретения в качестве экзогенной последовательности ДНК может быть использован участок двуцепочечной или одноцепочечной ДНК из генома организма, используемого при введении белка RbCpf1, но при этом измененный мутациями (заменой нуклеотидов), а также вставками, делециями, транслокациями, инверсиями одного или нескольких нуклеотидов.
Примеры
1. Нуклеаза RbCpf1 была выделена из бактерии Ruminococcus bromii из микробиома образцов кала человека. Для обнаружения нуклеазы проводили поиск белка (гипотетического или с неизвестной функцией). Для ДНК бактериальных геномов из нашей локальной базы данных был идентифицирован участок CRISPR-Cas, определенный как нуклеотидный локус, который содержал 25 т.п.н. последовательности геномной ДНК, фланкирующей массив CRISPR и гены белков cas. Открытые рамки считывания (ORF) в геномной ДНК были идентифицированы с помощью программы Prodigal (алгоритм поиска генов PROkaryotic DYnamic programming) (10.1186 / 1471-2105-11-119). Каждую ORF аннотировали с помощью поиска BLAST в базе данных non-redundant (nr) белков NCBI. Для обеспечения максимальной чувствительности обнаружения поиск проводился по модифицированным профилям последовательностей Cas1, Cas2, Cas4 и белку-эффектору в транслируемых геномных и метагеномных последовательностях (10.1038 / s41579-019-0299-x). Чтобы гарантировать, что массив CRISPR был обнаружен с высоким уровнем чувствительности, данные предсказаний, сделанные с использованием методов Piler-CR (10.1186 / 1471-2105-8-18) и CRISPRfinder (10.1093 / nar / gkm360), были объединены и использованы в качестве окончательного набора CRISPR. Участок CRISPR-Cas был извлечен, чтобы идентифицировать предсказанные гены, кодирующие белок, в непосредственной близости. Для гипотетических и ранее не идентифицированных последовательностей для предсказания доменной структуры использовали HHpred (10.1093 / nar / gki408). Алгоритм PSI-BLAST был использован для поиска отдаленных гомологов каждого обнаруженного белка (10.1093 / nar / 25.17.3389). Обнаруженные последовательности были отсортированы по следующим критериям: процент сходства с известными белками, размер (700-1400 аминокислот) и оптимальная температура для бактерий-хозяев. Этот алгоритм поиска помог нам сузить область поиска до последовательности из Ruminococcus sp. Сравнительный анализ последовательности и архитектуры белка показал, что нуклеаза из Ruminococcus bromii наиболее близка к семейству ферментов CRISPR-Cas V-A подтипа, которые содержат единственный нуклеазный домен RuvC, который отвечает за РНК-управляемое расщепление ДНК. Установлено, что размер гена нуклеазы RbCpf1 составляет 3736 пар нуклеотидов (b.p.).
Для получения последовательности гена белка-нуклеазы RbCpf1 использовали ПЦР (использовали 5’-фосфорилированные праймеры: прямой (5’-TGAACATATGATGCAAGAGCGTAAAAAAATATCGCATC-3’) и обратный (5’-ATCTACTCGAGATTATTCGCCATATCATTCTCCTGAACA-3’)). Полученные последовательности, содержащие ген белка RbCpf1, клонировали в линеаризованный вектор pUC118. Плазмидную ДНК трансформировали в штамм E. coli TOP10 и нарабатывали необходимое количество для секвенирования. Секвенирование проводили с помощью Illumina MiSeq. Ген RbCpf1 клонировали в вектор pET30a+ для трансформации экспрессионного штамма E. coli Rosetta2. Белок RbCpf1 выделяли и очищали из клеток E. coli Rosetta2 с использованием хроматографической системы GE AKTA Purifier 100 FPLC.
Для клеток человека впоследствии была проведена оптимизация кодонового состава гена RbCpf1, после чего химически синтезирован плазмидный вектор, кодирующий кодон-оптимизированный ген RbCpf1 (синтез выполнен компанией GenScript LCC).
Установлено, что нуклеаза RbCpf1 обладает сравнительно небольшим размером среди ортологов нуклеаз Cpf1, измеряемым в аминокислотных остатках (aa), что является несомненным преимуществом, так как размер является важной характеристикой для доставки нуклеаз внутрь клетки. Нуклеазы большего размера с доказанной эффективностью в клетках человека, в том числе RbCpf1:
MbCpf1 - 1373 aa
AsCpf1 - 1318 аа
HkCpf1 - 1310 aa
LpCpf1 - 1306 aa
TsCpf1 - 1306 aa
FnCpf1 - 1300 aa
CeCpf1 - 1286 aa
EeCpf1 - 1282 aa
ArCpf1 - 1263 aa
ErCpf1 - 1263 aa
Mb3Cpf1 - 1261 aa
Mb2Cpf1 - 1259 aa
RbCas12a - 1245 aa
Определение времени разрезания ДНК-матриц по сравнению с AsCpf1. Инкубация RbCpf1, crРНК и ДНК-матрицы, несущей протоспейсер, фланкированный последовательностью PAM, приводила к эффективному расщеплению RbCpf1 мишени в диапазоне от 1 секунды до 20 минут. Установлено, что RbCpf1 способен расщеплять субстрат ДНК, полученный с помощью ПЦР, всего за одну секунду после добавления нуклеазы в смесь для расщепления. Половина ДНК-субстрата расщепляется за 15 секунд для RbCpf1 и за 30 секунд для AsCas12a. Практически весь субстрат ДНК расщепляется обеими нуклеазами за 20 мин (фиг. 1).
In vitro разрезание проводили в объеме 30 мкл в буфере Cleavage Buffer, а также 1хNEB2 buffer (New England Biolabs Inc.). 10 нМ ДНК матрицы, 500 нМ нуклеазы, 5000 нМ crРНК инкубировали от 5 до 60 минут при температуре 37 градусов Цельсия. Реакцию останавливали добавлением Proteinase K (New England Biolabs Inc.) (1 единица активности) и инкубировали 15 минут при комнатной температуре. Анализировали результат разрезания с помощью электрофореза в 1% агарозном геле в 1хTAE буфере. Окрашенный EtBr гель помещали в Gel Doc EZ Imager (Bio-Rad) и оценивали с помощью программного обеспечения Image Lab Software 6.0.1 (Bio-Rad). Показано, что нуклеаза RbCpf1 проводит разрезание быстрее AsCpf1.
Матрица для in vitro разрезания представляла собой фрагменты ДНК, амплифицированные с помощью ПЦР в Q5® High-Fidelity 2X Master Mix (M0492L, NEB). В качестве матрицы использовали геномную ДНК, выделенную из линии клеток T-REx (линия клеток T-REx™ -293, Invitrogen, кат. № R71007) с помощью набора для очистки геномной ДНК GeneJET (K0722, Thermo Scientific™). Концентрацию ДНК измеряли флуорометром Qubit 3.0 с использованием набора Qubit DNA BR Assay Kit (Q32853, Thermo Scientific™).
2. Определение активности RbCpf1 в различных буферах при проведении реакций разрезания in vitro показало активность нуклеазы во всех тестированных буферах. Для этого смешивали рекомбинантный белок RbCpf1, соответствующую crРНК и линеаризованную ДНК-матрицу в виде PAM-библиотеки в соотношении 3:30:1 соответственно, с конечной концентрацией РНК 0.5 мМ, смесь далее инкубировали при 37 °C в течение 30 минут, после чего инактивировали нуклеазу добавлением 1 ед.акт. протеиназы К (New England Biolabs). В качестве буферов тестировали коммерчески-доступные буферы, упомянутые в работах по ортологичным ферментам: Nebbuffer1.1, Nebbuffer2.1, Nebbuffer3.1, Nebbuffer4, CutSmart, а также эти буферы с добавлением 5 мМ дитиотрейтола (ДТТ) и дополнительно буфер для разрезания (CB) (100 мМ HEPES pH 7.5; 500 мМ KCl; 25 мМ MgCl2; 5 мМ DTT). Эффективности разрезаний определяли с помощью программного обеспечения Bio-Rad ImageLab при подсчете интенсивностей полос на электрофореграммах (фиг. 2).
Матрица для in vitro разрезания представляла собой фрагменты ДНК, амплифицированные с помощью ПЦР в Q5® High-Fidelity 2X Master Mix (M0492L, NEB). В качестве матрицы использовали геномную ДНК, выделенную из линии клеток T-REx (линия клеток T-REx™-293, Invitrogen, кат. № R71007) с помощью набора для очистки геномной ДНК GeneJET (K0722, Thermo Scientific™). Концентрацию ДНК измеряли флуорометром Qubit 3.0 с использованием набора Qubit DNA BR Assay Kit (Q32853, Thermo Scientific™).
3. Нуклеаза RbCpf1 активна в диапазоне температур от 20 до 42 градусов Цельсия. В качестве стандартного буфера для разрезания in vitro брали Nebbuffer 2.1. Для определения температурного оптимума использовали точки в 15 °C, 20 °C, 25 °C, 30 °C, 37 °C, 42 °C, 48 °C. Соотношения ДНК-матрицы:РНК:RbCpf1 использовали 1:30:3. Фрагменты анализировали электрофоретически в 1х ТАЕ 1 % агарозном геле с добавлением этидия бромида (фиг. 3).
Матрица для in vitro разрезания представляла собой фрагменты ДНК, амплифицированные с помощью ПЦР в Q5® High-Fidelity 2X Master Mix (M0492L, NEB). В качестве матрицы использовали геномную ДНК, выделенную из линии клеток T-REx (линия клеток T-REx™ -293, Invitrogen, кат. № R71007) с помощью набора для очистки геномной ДНК GeneJET (K0722, Thermo Scientific™). Концентрацию ДНК измеряли флуорометром Qubit 3.0 с использованием набора Qubit DNA BR Assay Kit (Q32853, Thermo Scientific™).
4. Определяли наиболее частый контекст РАМ, при опознавании которого происходило разрезание. Установлено, что оптимальным является PAM, содержащий 5’-YYN-3’, либо представленный только 5’-YYN-3. Для поиска РАМ проверяли способность RbCpf1 расщеплять линейные библиотеки PAM ДНК, содержащие сайт-мишень, фланкированный восемью рандомизированными нуклеотидами на 5’-конце. RbCas12a в комплексе с crРНК инкубировали с библиотекой PAM при 37 °С в течение 1 часа, расщепленные и нерасщепленные молекулы очищали после электрофореза в агарозном геле и секвенировали с использованием платформы Illumina. Биоинформатический анализ в программном обеспечении WebLogo3.7.4 позволил определить PAM. Результаты показали, что RbCpf1 предпочитает мишени, примыкающие к 5’-YYN-3’ PAM (см. фиг. 4).
В качестве матриц в независимых реакциях ПЦР (ДНК-полимераза Q5, NEB) использовали два M13-фланкированных обратно-комплементарных олигонуклеотида размером 100 п.н. с вырожденными фрагментами длиной 8 п.о. (Синтол, Россия), примыкающими к протоспейсеру. Ампликоны анализировали с помощью электрофореза в агарозном геле с последующей экстракцией из геля (Qiagen Gel Extraction Kit). Проводили рестрикцию полученных фрагментов с помощью EaeI (NEB) и клонировали в вектор pBR322, линеаризованный с помощью EagI и PvuII (NEB), с последующей трансформацией в штамм E.coli Top10. Через шестнадцать часов после трансформации с планшетов было смыто более 180 000 колоний, и библиотека плазмид была экстрагирована с помощью GenElute™ HP Plasmid Maxiprep Kit (Sigma). Последовательности используемых праймеров: прямой - GTAAAACGACGGCCAGTgccgcagtactgatcatNNNNNNNNcccctctattgatccccacctccaaatatctcatcaacaacGTCATAGCTGTTTCCTG, обратный - CAGGAAACAGCTATGACgttgttgatgagatatttggaggtggggatcaatagaggggNNNNNNNNatgatcagtactgcggcACTGGCCGTCGTTTTAC.
5. Результаты фиг. 4 подтверждали in vitro в два этапа. Для этого на первом этапе получили синтетические конструкции, содержащие один протоспейсер, но разные 3'-концевые нуклеотиды в последовательности PAM 5-TTN-3. Используя пары праймеров, вносящих мутации в третий нуклеотид PAM, и набор Q5® Site-Directed Mutagenesis Kit, получили 4 генетические конструкции, содержащие один протоспейсер и один из четырех PAM: 5'-TTC-3', 5'-TTG-3', 5'-TTA-3', 5'-TTT-3'. Полученные векторы линеаризовали ScaI рестриктазой (New England Biolabs) и гидролизовали с использованием RbCpf1 и направляющей РНК. Реакции проводили в трех повторах. Результаты гидролиза анализировали электрофоретически в 1х ТАЕ 1 % агарозе с окраской этидием бромидом. Чтобы подтвердить данные, полученные после NGS и отображенные на фиг. 4, на втором этапе к плазмидному вектору pUC119 были подобраны 13 crРНК на 5'-YYN-3' PAM: 12 РНК на 5'-CTN-3' и 5'-TCN-3' PAM, и одна дополнительная на 5'-TTC-3' PAM в качестве контроля. Используя полученные crРНК, ставили реакции разрезания in vitro с RbCas12a и HindIII рестриктазой (New England Biolabs) в трех. Результаты гидролиза анализировали электрофоретически в 1х ТАЕ 1 % агарозе с окраской этидием бромидом. Результаты представлены на фиг. 5.
6. Нуклеазу испытывали в клетках эукариот на примере человеческой клеточной линии HEK293T. Результаты представлены на фиг. 6. Кодон-оптимизированный ген RbCas12a клонировали в плазмидный вектор под регуляцией промотора CMV. Последовательности, кодирующие последовательности crРНК, клонировали в независимые плазмидные векторы под контролем промотора U6 (фиг. 17). RbCpf1 с помощью двух направляющих РНК нацеливали на человеческий ген DNMT1.
Для определения разрезания в клетках эукариот клеточная линия HEK293T культивировалась в среде DMEM с добавлением 10% FBS при 37 градусах Цельсия и 5 % углекислоты. Смена среды на OptiMEM производилась за час до трансфекции. Трансфекция производилась в 6-луночном планшете с помощью реагента Lipofectamine3000 (ThermoScientific). Трансфецировали 2000 нг плазмид, кодирующих RbCpf1 и две направляющих РНК. Через 48 часов клетки снимали и производили выделение ДНК с помощью набора Qiamp DNA MINI KIT (Qiagen). Далее амплифицировали участок, содержащий ген DNMT1, с помощью фосфорилированных праймеров, анализировали с помощью гель-электрофореза и проводили очистку фрагментов ДНК из агарозного геля. Выделенные фрагменты клонировали в составе плазмидного вектора pUC119 и секвенировали по Сэнгеру.
7. Нуклеазу испытывали в условиях in vitro, где в качестве белка Cpf1 использовали рекомбинантные препараты с 97 % (RbCpf1_mut и RbCpf1_mut2) гомологии с SEQ ID NO:1. Результаты разрезаний приведены на фиг. 13. Мутации в последовательностях RbCpf1_mut и RbCpf1_mut2 обозначены на фиг. 14-15.
| название | год | авторы | номер документа |
|---|---|---|---|
| Средство разрезания двунитевой ДНК с помощью Cas12d белка из Katanobacteria и гибридной РНК, полученной путем слияния направляющей CRISPR РНК и scout РНК | 2020 |
|
RU2771626C1 |
| СРЕДСТВО РАЗРЕЗАНИЯ ДНК НА ОСНОВЕ CAS9 БЕЛКА ИЗ DEFLUVIIMONAS SP. | 2018 |
|
RU2712492C1 |
| Средство разрезания ДНК на основе Cas9 белка из биотехнологически значимой бактерии Clostridium cellulolyticum | 2018 |
|
RU2712497C1 |
| Средство разрезания ДНК на основе ScCas12a белка из бактерии Sedimentisphaera cyanobacteriorum | 2022 |
|
RU2791447C1 |
| ГИБРИДНЫЕ ДНК/РНК-ПОЛИНУКЛЕОТИДЫ CRISPR И СПОСОБЫ ПРИМЕНЕНИЯ | 2016 |
|
RU2713328C2 |
| СРЕДСТВО РАЗРЕЗАНИЯ ДНК НА ОСНОВЕ CAS9 БЕЛКА ИЗ БАКТЕРИИ DEMEQUINA SEDIMINICOLA | 2019 |
|
RU2722933C1 |
| Средство разрезания ДНК на основе Cas9 белка из бактерии Streptococcus uberis NCTC3858 | 2022 |
|
RU2788197C1 |
| ПРИМЕНЕНИЕ CAS9 БЕЛКА ИЗ БАКТЕРИИ PASTEURELLA PNEUMOTROPICA ДЛЯ МОДИФИКАЦИИ ГЕНОМНОЙ ДНК В КЛЕТКАХ | 2019 |
|
RU2724470C1 |
| СИСТЕМА РЕДАКТИРОВАНИЯ ГЕНОМНОЙ ДНК ЭУКАРИОТИЧЕСКОЙ КЛЕТКИ НА ОСНОВЕ НУКЛЕОТИДНОЙ ПОСЛЕДОВАТЕЛЬНОСТИ, КОДИРУЮЩЕЙ БЕЛОК SUCAS9NLS | 2022 |
|
RU2804422C1 |
| СРЕДСТВО РАЗРЕЗАНИЯ ДНК НА ОСНОВЕ CAS9 БЕЛКА ИЗ БАКТЕРИИ PASTEURELLA PNEUMOTROPICA | 2019 |
|
RU2722934C1 |








Настоящее изобретение относится к области биотехнологии, в частности к ферменту нуклеазе и применению данного фермента. Изобретение относится к ферменту нуклеазе Cpf1 из бактерии Ruminococcus bromii и молекуле ДНК, кодирующей указанный фермент. Настоящее изобретение также относится к вектору, содержащему указанную молекулу ДНК, системе CRISPR/Cpf1, содержащей указанную нуклеазу и направляющую РНК, клетке-хозяину для получения нуклеазы Cpf1, способу получения нуклеазы Cpf1, применению указанной нуклеазы Cpf1 для образования разрыва в молекуле ДНК или РНК, способу образования разрыва в молекуле ДНК или РНК с использованием указанной нуклеазы, системе доставки, содержащей указанную нуклеазу Cpf1, молекулу или вектор, комбинации способов доставки, содержащих указанную нуклеазу Cpf1, молекулу ДНК или вектор, способу модификации целевого локуса ДНК с использованием указанной нуклеазы, применению указанной нуклеазы Cpf1 для редактирования генома или генной терапии и детекции нуклеиновых кислот. Изобретение позволяет расширить пул доступных нуклеаз для геномного редактирования. 8 н. и 13 з.п. ф-лы, 15 ил., 1 пр.
1. Белок, обладающий активностью нуклеазы, характеризующийся тем, что содержит аминокислотную последовательность SEQ ID NO: 1.
2. Белок по п. 1, характеризующийся тем, что представляет собой природный белок, или искусственно синтезированный белок, или рекомбинантный белок, или белок, полученный в бесклеточной системе трансляции, или искусственно гибридный белок.
3. Молекула нуклеиновой кислоты, кодирующая белок по п. 1, характеризующаяся тем, что содержит нуклеотидную последовательность SEQ ID NO: 2 или последовательность, которая по меньшей мере на 98% идентична нуклеотидной последовательности SEQ ID NO: 2.
4. Вектор экспрессии, содержащий молекулу нуклеиновой кислоты по п. 3 и элементы, обеспечивающие поддержание целостности и/или копийности и/или функциональности вектора.
5. Вектор по п. 4, характеризующийся тем, что содержит элементы, обеспечивающие поддержание целостности и/или копийности и/или функциональности молекулы ДНК, включающие ориджины репликации, гены устойчивости к антибиотикам, промоторные, операторные, энхансерные и терминаторные регионы генов, регионы связывания транскрипционных факторов.
6. Вектор по п. 4, характеризующийся тем, что вектор для экспрессии RbCpf1 в клетках человека включает следующие последовательно расположенные элементы:
Энхансер цитомегаловируса (CMV)-235-614 п.о.
Промотор цитомегаловируса (CMV)-615-818 п.о.
T7 промотор-863-881 п.о.
Ген RbCpf1-917-4651 п.о.
Сигнал ядерной локализации нуклеоплазмина NLS-4652-4699 п.о.
Аффинная метка гемаглютинина вируса гриппа HA-4700-4726 п.о.
Сигнал полиаденилирования бычьего гормона роста bGH-poly(A)-4832-5056 п.о.
Ориджин репликации f1-5102-5530 п.о.
Промотор и энхансер обезьяньего вируса SV40-5544-5873 п.о.
Ориджин репликации обезьяньего вируса SV40-5724-5859 п.о.
Ген устойчивости к канамицину, неомицину, G418 NeoR/KanR-5940-6734 п.о.
Сигнал полиаденилирования обезьяньего вируса SV40-6908-7029 п.о.
Lac оператор-7102-7118 п.о.
Промотор lac-7126-7156 п.о.
Сайт связывания CAP-717-7192 п.о.
Ориджин репликации ColE1/pMB1/pBR322/pUC-7480-8065 п.о.
Ген устойчивости к ампициллину AmpR-8236-9096 п.о.
Промотор гена AmpR-9097-9201 п.о.
7. Вектор по п. 4, характеризующийся тем, что является вектором для экспрессии в клетках Escherichia coli, включает следующие последовательно расположенные элементы:
T7 терминатор-26-73 п.о
6xHis аффинная метка-140-157 п.о.
Ген RbCpf1-159-3898 п.о.
Сайт связывания рибосом из гена 10 фага T7 (RBS)-3909-3931 п.о.
Lac оператор-3946-3970 п.о.
T7 промотор-3971-3989 п.о.
lacI промотор-4302-4379 п.о.
Ген lacI-4380-5462 п.о.
Ген rop-6271-6462 п.о.
bom элемент-6564-6706 п.о.
Ориджин репликации ColE1/pMB1/pBR322/pUC-6892-7480 п.о.
Ген устойчивости к канамицину KanR-7602-8417 п.о.
Ориджин репликации фага f1-8510-8965 п.о.
8. Клетка-хозяин для продуцирования белка по п. 1, характеризующаяся тем, что содержит молекулу нуклеиновой кислоты по п. 3 или вектор по п. 4.
9. Клетка-хозяин по п. 8, характеризующаяся тем, что представляет собой прокариотическую или эукариотическую клетку.
10. Клетка-хозяин по п. 9, характеризующаяся тем, что клетка-хозяин представляет собой клетку бактерии, гриба, растения или животного, насекомого или млекопитающего.
11. Клетка-хозяин по п. 10, характеризующаяся тем, что представляет собой клетку организма, выбранного из родов, включающих Escherichia, Bacillus, Saccharomyces, Schizosaccharomyces, Pichia, Synechocystis, Chlamydomonas, Anabaena, Arabidopsis, Solanum, Triticum, Nicotiana, Hordeum, Drosophila, Mus, Rattus, Homo.
12. Способ получения белка по п. 1, характеризующийся тем, что включает этапы культивирования клетки-хозяина по п. 8 в культуральной среде, подходящей для культивирования клетки-хозяина в условиях, обеспечивающих продуцирование белка.
13. Способ по п. 12, характеризующийся тем, что включает этапы выделения и/или очистки продуцированного белка.
14. Система CRISPR/Cpf1, характеризующаяся тем, что включает направляющую (crispr) РНК для белка и содержащая белок по п. 1.
15. Применение белка по п. 1 для образования разрыва в молекуле нуклеиновой кислоты.
16. Применение по п. 15, характеризующееся тем, что разрыв может быть однонитевым или двунитевым.
17. Применение по п. 15, характеризующееся тем, что молекула нуклеиновой кислоты представляет собой ДНК или РНК.
18. Система доставки нуклеазы в живые клетки, характеризующаяся тем, что включает белок по п. 1, молекулу нуклеиновой кислоты по п. 3 или вектор по п. 4, при этом система содержит элементы, обеспечивающие поддержание целостности и/или копийности и/или функциональности молекулы ДНК, включающие ориджины репликации, гены устойчивости к антибиотикам, промоторные, операторные, энхансерные и терминаторные регионы генов, регионы связывания транскрипционных факторов.
19. Применение по п. 15, характеризующееся тем, что образование разрывов осуществляют для редактирования генома одноклеточного или многоклеточного организма, или для коррекции генетического дефекта в виде геномных мутаций в организме, включающее внесение в по меньшей мере одну клетку указанного организма белка по п. 1, или молекулы ДНК или РНК по п. 3, или вектора по п. 4 или системы CRISPR/Cpf1 по п. 14.
20. Применение по п. 19, характеризующееся тем, что организм представляет собой прокариотический или эукариотический организм, предпочтительно простейшее, бактерию, растение, грибы, животное, предпочтительно млекопитающее, в частности человек.
21. Применение по п. 15, характеризующееся тем, что коррекцию генетического дефекта осуществляют путем внесения однонитевых или двунитевых разрывов в молекулы ДНК в присутствии или без такового донорной последовательности или в виде нарушений экспрессии генов, таких как снижение или повышение экспрессии генов, путем направленного изменения экспрессии, опосредованного использованием белка Cpf1 по п. 1, молекулы ДНК или РНК по п. 3, или вектора по п. 4, или системы CRISPR/Cpf1 по п. 14.
| Гальванометрическое реле | 1942 |
|
SU72854A1 |
| Новая компактная нуклеаза CAS9 II типа из Anoxybacillus flavithermus | 2020 |
|
RU2749307C1 |
| ГЕНЕТИЧЕСКАЯ КОНСТРУКЦИЯ НА ОСНОВЕ СИСТЕМЫ РЕДАКТИРОВАНИЯ ГЕНОМА CRISPR/CAS9, КОДИРУЮЩАЯ НУКЛЕАЗУ CAS9, ДЛЯ ГЕНОМНОГО РЕДАКТИРОВАНИЯ ОДНОДОЛЬНЫХ ЗЕРНОВЫХ КУЛЬТУР НА ОСНОВЕ ДВОЙНОГО ОТБОРА РАСТЕНИЙ | 2020 |
|
RU2762830C1 |
| СРЕДСТВО РАЗРЕЗАНИЯ ДНК НА ОСНОВЕ CAS9 БЕЛКА ИЗ БАКТЕРИИ PASTEURELLA PNEUMOTROPICA | 2019 |
|
RU2722934C1 |
| WO 2017048969 A1, 23.03.2017 | |||
| WO 2015006294 A2, 15.01.2015 | |||
| СРЕДСТВО РАЗРЕЗАНИЯ ДНК НА ОСНОВЕ CAS9 БЕЛКА ИЗ DEFLUVIIMONAS SP. | 2018 |
|
RU2712492C1 |
Авторы
Даты
2024-04-08—Публикация
2022-12-30—Подача