Родственные заявки
В настоящей заявке испрашивается право на приоритет заявок на патент Китая N№ 201010213717.6, 201010213719.5 и 201010213721.2, поданных 30 июня 2010 г., и право на приоритет Международных заявок N№ PCT/CN2010/002150 и PCT/CN2010/002149, поданных 24 декабря 2010 г., содержание которых во всей своей полноте вводится в настоящее описание посредством ссылки.
Область техники, к которой относится изобретения
Изобретение относится к области секвенирования нуклеиновых кислот, в частности к области ПЦР-секвенирования. Настоящее изобретение также относится к технике считывания штрихового кода ДНК и к стратегии неполного фрагментирования ДНК. Способ согласно изобретению может быть применен, в частности, в технологии секвенирования второго поколения, а особенно, в технологии парно-концевого секвенирования второго поколения, а также в генотипировании HLA. Более конкретно, настоящее изобретение относится к способу генотипирования HLA, а в частности, к способу генотипироавания HLA-A, HLA-B, HLA-C и HLA-DQB1, а также к парам праймеров, используемым в ПЦР-амплификации, применяемой в этом способе.
Предшествующий уровень техники
Метод ПЦР-секвенирования означает технологию, в которой ДНК-фрагменты представляющего интерес гена получают методом ПЦР, а затем полученные ДНК-фрагменты представляющего интерес гена подвергают ДНК-секвенированию с получением информации о последовательности ДНК представляющего интерес гена. Методы ПЦР-секвенирования широко применяются в таких областях, как детектирование мутации генов и генотипирование в течение длительного периода времени.
Технология ДНК-секвенирования подразделяется, главным образом, на технологию ДНК-секвенирования первого поколения, осуществляемую методом секвенирования по Сэнгеру, и на технологию ДНК-секвенирования второго поколения, осуществляемую на секвенаторе Illumina GA, Roche 454, ABI Solid, и т.п. Метод ДНК-секвенирования по Сэнгеру отличается простотой экспериментальных операций с получением визуальных и точных результатов и непродолжительностью времени проведения эксперимента, а поэтому этот метод широко применяется в таких областях, как клиническое детектирование мутации генов и генотипирование, где необходима быстрая оборачиваемость процесса, особенно, если это касается результатов детектирования. Однако из-за низкой производительности и высокой стоимости этого метода, его применение в области крупномасштабного генотипирования имеет определенные ограничения.
По сравнению с технологией ДНК-секвенирования первого поколения, технология ДНК-секвенирования второго поколения отличается тем, что она имеет высокую производительность секвенирования, низкую стоимость, высокий уровень автоматизации и возможность одномолекулярного секвенирования. Если взять в качестве примера технологию одномолекулярного секвенирования Illumina GA, то путем секвенирования по одной молекуле можно получить данные для 50G (около 50 миллиардов) оснований, то есть в среднем 5 миллиардов оснований в день, при этом средняя стоимость секвенирования основания составляет менее чем 1/1000 от стоимости секвенирования по методу Сэнгера. Кроме того, анализ результатов может быть непосредственно осуществлен на компьютере. Таким образом, технология секвенирования ДНК на секвенаторе второго поколения представляет собой технологию, наиболее подходящую для крупномасштабного секвенирования. Однако в технологии ДНК-секвенирования второго поколения, длина участка непрерывного секвенирования по всей его длине обычно невелика. В настоящее время максимальная длина двунаправленного секвенирования на секвенаторе Illumina GA составляет 200 п.н., и хотя в методе Roche 454 GS-FLX максимальная длина последовательности для секвенирования может составлять примерно до 500 п.н., однако затраты на такое секвенирование довольно велики, а производительность - низка. Если ПЦР-ампликон имеет длину, превышающую максимальную длину, которая может быть непрерывно секвенирована в данном секвенаторе, то секвенирование всего ампликона не может быть осуществлено путем прямого секвенирования, а поэтому не может быть получена информация о всей длине последовательности ДНК ампликона. Из-за короткой максимальной длины секвенирования применение технологии секвенирования второго поколения в методе ПЦР-секвенирования имеет определенные ограничения. Помимо поэтапного усовершенствования технологии секвенирования в целях увеличения максимальной длины секвенирования, крайне необходимо разработать новый способ, который позволял бы решить существующие в настоящее время проблемы, связанные с максимальной длиной ДНК-секвенирования на секвенаторе второго поколения, и который можно было бы применять в технологии ПЦР-секвенирования.
Главный комплекс гистосовместимости человека (HLA) представляет собой одну из генных систем, которые, как известно, имеют самый высокий полиморфизм. HLA представляет собой главную генную систему для модуляции специфического иммунного ответа в организме человека и определения различий в восприимчивости индивидуумов к развитию заболеваний и непосредственно ассоциируется с отторжением аллогенных органов-трансплантатов. Проведенные исследования показали, что чем выше степень соответствия генов, таких как HLA-A, B, C, DRB1 и DQB1, а также степень их разрешения при генотипировании у донора и реципиента, тем выше выживаемость трансплантата. В настоящее время перед трансплантацией гемопоэтических стволовых клеток уже проводится стандартная процедура тестирования, заключающаяся в высокопроизводительном генотипировании HLA у потенциальных доноров и реципиентов.
Современная международная стандартная технология высокоразрешающего HLA-генотипирования представляет собой метод ПЦР-секвенирования, проводимый на основе технологии секвенирования по Сэнгеру, которая включает ПЦР-амплификацию соответствующих областей гена HLA, секвенирование амплифицированного продукта, генотипирование результатов секвенирования с помощью профессиональной компьютерной программы по генотипированию и, наконец, получение информации о генотипе HLA-образца. Этот метод отличается тем, что он дает визуальные результаты, имеет высокое разрешение и позволяет детектировать новые аллели. Однако из-за высокой стоимости и низкой производительности метода секвенирования по Сэнгеру, его применение в научно-исследовательских институтах, имеющих регистрационную базу данных о добровольцах-донорах гемопоэтических стволовых клеток (банк клеток костного мозга), где требуется проведение крупномасштабного детектирования путем HLA-генотипирования, имеет определенные ограничения.
Сообщалось, что для HLA-генотипирования было применено ПЦР-секвенирование методом Roche 454 GS-FLX. Однако поскольку этот метод является довольно дорогостоящим, то он не имеет явных преимуществ в отношении производительности и стоимости секвенирования по сравнению с методом HLA-генотипирования, проводимым путем секвенирования по Сэнгеру. По сравнению с методом Roche 454 GS-FLX, метод Illumina GA оперирует с более короткой максимальной длиной непрерывного секвенирования, но имеет явные преимущества, такие как высокая производительность и низкая стоимость секвенирования. Если будет решена проблема, связанная с небольшой максимальной длиной секвенирования в методе Illumina GA, то применение этого метода для HLA-генотипирования позволит скомпенсировать недостатки современных методов HLA-генотипирования.
Описание сущности изобретения
При одновременном проведении анализов путем секвенирования последовательностей со специфическим геном в большом числе образцов с применением технологии секвенирования второго поколения обычно применяется стратегия ПЦР-секвенирования, в которой непосредственно используется комбинация индексирования праймера и технологии секвенирования второго поколения. Если максимально возможная длина непрерывного секвенирования в секвенаторе может покрывать длину всего ПЦР-продукта, то вышеуказанная стратегия удовлетворяет предъявляемым требованиям. Если максимальная длина секвенирования в секвенаторе не может покрывать длину всего ПЦР-продукта, то секвенатор Illumina GA необходимо заменить секвенатором второго поколения, имеющим большую максимальную длину секвенирования (таким как Roche 454 GS-FLX). Если максимальная длина секвенирования последовательности все еще не удовлетворяет этим требованиям, то может быть использован секвенатор первого поколения со скарификацией стоимости и производительности.
Фактически, ситуация заключается в том, что секвенатор Illumina GA отличается исключительно высокой производительностью, но его максимальная длина секвенирования составляет лишь 200 п.н.; и хотя максимальная длина секвенирования на секвенаторе Roche 454 GS-FLX может достигать примерно 500 п.н., но стоимость такого секвенирования остается относительно высокой, а производительность - относительно низкой; и кроме того, хотя максимальная длина секвенирования на секвенаторе первого поколения может достигать более чем 1000 п.н., однако его производительность и стоимость не сравнима с производительностью и стоимостью секвенатора второго поколения.
Авторам пока не известен метод, позволяющий увеличивать длину ПЦР-продуктов, которые могут быть секвенированы по всей их длине без скарификации стоимости и производительности. Комбинация индексирования праймеров, стратегии неполного фрагментирования ДНК и применения технологии секвенирования второго поколения, описанных в настоящей заявке, позволяет оперировать с ПЦР-продуктами, которые имеют длину, превышающую максимальную длину непрерывного секвенирования на секвенаторе, и при этом позволяет полностью использовать преимущества технологии секвенирования второго поколения, такие как высокая производительность и низкая стоимость, что значительно расширяет область применения такой технологии. Среди прочих технологий секвенирования второго поколения, технология секвенирования второго поколения, применяемая в настоящем изобретении, включает секвенирование с обоих концов и ПЦР-секвенирование, в котором для ПЦР-матрицы используется эталонная последовательность ДНК.
Настоящее изобретение относится к способам ПЦР-секвенирования, которые позволяют устранить ограничения, связанные с короткой максимальной длиной секвенирования на секвенаторе и расширить область применения технологии второго поколения в ПЦР-секвенировании ДНК. Так, например, при секвенировании с применением технологии второго поколения используют индексированные праймеры, имеющие индекс, добавленный к его 5'-концу, и амплифицированные ПЦР-продукты фрагментируют, после чего фрагментированные продукты репарируют по концам, и к их 3'-концам присоединяют дезоксиаденозин (A), а затем лигируют с различными адаптерами, полученными без проведения ПЦР.
Метод ПЦР-секвенирования, проводимый на основе штрихового кодирования ДНК и стратегии неполного фрагментирования ДНК, позволяет значительно увеличить число специфически меченых образцов без увеличения числа индексов праймера (фигура 5). В настоящем изобретении фактически секвенируемая длина ПЦР-продуктов превышает максимальную длину секвенирования на секвенаторе из-за добавления индексов праймера к прямому и обратному ПЦР-праймерам, и секвенирование осуществляют в комбинации со стратегией неполного фрагментирования ДНК и с технологией секвенирования второго поколения.
Добавление последовательности-индекса к переднему концу праймера для амплификации осуществляют в целях реализации одновременного секвенирования множества образцов. Более конкретно, уникальный индекс праймера добавляют к каждому образцу в процессе ПЦР с применением технологии ПЦР-индекса/штрих-кода в комбинации с синтезом индексированного праймера путем добавления индекса к 5'-концу ПЦР-праймера. Так, например, во время секвенирования, проводимого с применением технологии секвенирования второго поколения, образцы последовательно процессируют только в стадии ПЦР, а в остальных экспериментальных стадиях эти образцы могут быть смешаны и процессированы одновременно, и конечный результат для каждого образца может быть идентифицирован по индексу его уникального праймера.
Метод индексирования «адаптера» или «адаптера библиотеки» означает метод индексирования библиотек, включающий добавление к множеству библиотек для секвенирования различных адаптеров библиотек (различных адаптеров библиотек, состоящих из различных последовательностей и различных частей последовательностей, называемых адаптерными индексами), конструирование индексированных библиотек для секвенирования, и последующее секвенирование множества различных индексированных библиотек для секвенирования в пуле, где конечный результат секвенирования каждой индексированной библиотеки для секвенирования отличается друг от друга. Термин «адаптер PCR-FREE-библиотеки» означает сегмент, сконструированный из оснований, главная роль которых заключается в дополнительной фиксации молекулы ДНК на чипе для секвенирования и в обеспечении сайтов связывания для универсальных секвенирующих праймеров, где адаптер PCR-FREE-библиотеки может быть непосредственно лигирован с двумя концами ДНК-фрагментов в библиотеке для секвенирования. Поскольку при введении адаптера не проводят ПЦР, то такой адаптер называется здесь «адаптер PCR-FREE-библиотеки». Так, например, адаптеры PCR-FREE-библиотеки, используемые в примерах настоящего изобретения, были получены от ILLUMIA.
Метод конструирования PCR-FREE-библиотеки, где используется технология индексирования адаптера библиотеки, заключается в непосредственном лигировании адаптера библиотеки к двум концам ДНК-фрагмента в библиотеке для секвенирования. Поскольку при введении адаптера библиотеки не проводят ПЦР, то такой адаптер называется здесь «адаптер PCR-FREE-библиотеки». Для лигирования в процессе введения может быть использована ДНК-лигаза. Поскольку в процессе конструирования библиотеки не проводят ПЦР, то неточности конечных результатов ПЦР можно избежать во время конструирования библиотеки, содержащей ПЦР-продукты с большой степенью сходства последовательностей.
Методами амплификации ДНК, методами экстракции ДНК, методами очистки ДНК и методами выравнивания последовательностей ДНК, применяемыми в настоящем изобретении, могут быть методы, известные специалистам. Указанные методы могут быть выбраны самим специалистом, исходя из практических соображений. Что касается методов секвенирования ДНК, то специалист в данной области может осуществить их в соответствии со стандартными процедурами или в соответствии с инструкцией, прилагаемой к секвенатору.
Конструирование индексов праймера может быть осуществлено в зависимости от применяемой концепции эксперимента. Что касается концепции секвенирования методом Illumina GA, то в настоящем изобретении при конструировании праймерных индексов были предприняты следующие действия: 1) последовательность мононуклеотидного повтора, содержащая 3 или более основания, была удалена в последовательностях праймерных индексов, 2) общее количество оснований A и оснований C в одном и том же сайте всех праймерных индексов составляет 30%-70% от количества всех оснований, 3) содержание GC в последовательности праймерных индексов составляет 40-60%, 4) праймерные индексы отличаются друг от друга по меньшей мере 4 основаниями, 5) последовательности, имеющие высокую степень сходства с праймерами для секвенирования методом Illumina GA, были удалены из последовательностей-индексов праймеров, и 6) случаи, в которых добавление последовательностей праймерных индексов к ПЦР-праймерам приводит к образованию крупных шпилек и димеров, были сведены к минимуму.
В настоящем изобретении два праймерных индекса (которые являются либо идентичными, либо различными) добавляли к двум концам ПЦР-продукта, соответственно, так, чтобы индекс праймера у любого конца ПЦР-продукта мог служить специфической меткой, идентифицирующей образец ПЦР-продукта. Полученный ПЦР-продукт подвергали неполному фрагментированию. Так называемое «неполное фрагментирование» означает процесс получения продуктов, содержащих интактные нефрагментированные ПЦР-продукты и частично фрагментированные ПЦР-продукты. Методами фрагментирования являются, но не ограничиваются ими, методы химического фрагментирования (такие как ферментативное расщепление) и методы физического фрагментирования. Методами физического фрагментирования являются методы ультразвукового фрагментирования или методы механического фрагментирования. Фрагментированную ДНК подвергают электрофорезу в 2% агарозе, и все ДНК-полосы, имеющие длину между максимальной длиной секвенирования и максимально приемлемой длиной ДНК для секвенирования на секвенаторе, очищают и выделяют путем разрезания геля (наибольшая длина ДНК, приемлемая для секвенатора Illumina GA составляет 700 п.н., и эта длина означает исходную длину ДНК, которая не включает длину последовательности адаптера библиотеки). Методами очистки и выделения являются, но не ограничиваются ими, выделение путем электрофореза и разрезания геля, и выделение на магнитных сферах. Из выделенных ДНК-фрагментов конструируют библиотеки для секвенирования в соответствии с процедурами конструирования библиотек для секвенирования на секвенаторе второго поколения, а затем продукты подвергают секвенированию. Библиотеки для секвенирования предпочтительно конструируют в соответствии с PCR-FREE-процедурами, подходящими для конструирования библиотек для секвенирования, а методом секвенирования является метод секвенирования с обоих концов. Конструирование PCR-FREE-библиотек для секвенирования осуществляют методами, известными специалистам. При получении данных секвенирования, информация о последовательностях всех тест-образцов может быть получена исходя из последовательностей праймерных индексов. Считанные последовательности (последовательности-«риды») сопоставляют путем выравнивания с соответствующими эталонными ДНК-последовательностями ПЦР-продуктов с помощью BMA, а затем осуществляют сборку полноразмерной последовательности исходя из перекрывания и характера сцепления между последовательностями-ридами (фигура 1). Используемый здесь термин «сцепление» означает присоединение по концам, определяемое параметрами парно-концевого секвенирования.
Секвенирование на секвенаторе Illumina GA (геномный аналитический секвенатор, выпускаемый фирмой Illumina Inc., для краткости называемый здесь Illumina GA), то есть анализ последовательности ДНК, осуществляют, как в принципе и всякое секвенирование, путем синтеза. Этот анализ может быть применен в фазе гаплотипирования, и полученные данные, в конечном счете, представляют собой серию последовательностей оснований, которые могут быть непосредственно подвергнуты выравниванию с эталонными последовательностями, имеющимися в базе данных HLA. Поскольку эта программа не давала каких-либо заметных ошибок, а именно, несоответствий пиков, обычно наблюдаемых у существующих традиционных программ типирования, то желательно, чтобы эта программа типирования была автоматизирована. Секвенатор Illumina GA имеет высокую производительность секвенирования. В настоящее время один единственный раунд секвенирования позволяет получить данные о 50G (50 миллиардов) оснований, то есть, в среднем, 5 миллиардов оснований в день. Благодаря такой высокой производительности, для каждой последовательности может быть достигнута высокая процедурная мощность секвенирования, что дает гарантию надежности результатов секвенирования.
До сих пор каких-либо исследований по применению секвенирования на Illumina GA для HLA-типирования не проводилось. В настоящем изобретении впервые применяется секвенирование на Illumina GA для HLA-типирования, и такой метод HLA-типирования является не дорогостоящим и высокопроизводительным, а также имеет высокую точность и высокое разрешение, если его проводят в комбинации с применением технологии ПЦР-секвенирования и технологии считывания штрих-кода ДНК, с неполным фрагментированием ДНК и созданием PCR-FREE-библиотеки.
В настоящем изобретении с помощью метода ПЦР-секвенирования, основанного на технологии считывании штрих-кода ДНК, неполного фрагментирования ДНК и получения PCR-FREE-библиотеки, анализируемые образцы подразделяют на группы, после чего образцы каждой группы подвергают амплификации фрагментов представляющих интерес генов HLA с использованием праймеров, меченных двунаправленными праймерными индексами (максимальная длина ПЦР-продуктов зависит от максимальной длины ДНК, которая может быть использована в секвенаторе; при этом в современном секвенаторе Illumina GA максимально приемлемая длина считывания ДНК составляет 700 п.н. и такую длину имеет исходная ДНК, которая не включает последовательности адаптера библиотеки), затем ПЦР-продукты объединяют вместе в том же количестве и подвергают неполному фрагментированию с получением индексированной PCR-FREE-библиотеки для секвенирования ДНК. Различные индексированные библиотеки для секвенирования, полученные из образцов различных групп, смешивают в равном молярном объеме, а затем все ДНК-фрагменты, длина которых превышает максимальную длину секвенирования в секвенаторе, селективно выделяют и подвергают секвенированию на секвенаторе Illumina GA. Последовательности-риды ДНК для каждого образца могут быть получены путем скрининга информации о последовательностях индексов адаптеров, индексов праймеров и ПЦР-праймеров в общих данных по секвенированию. Полученные последовательности ДНК, после их сборки, сопоставляют с соответствующими данными, имеющимися в профессиональной базе данных HLA IMGT, и наконец, определяют генотип HLA образца.
В вышеописанных методах, после фрагментирования указанной ДНК, ДНК образцов различных групп лигируют с другим адаптером библиотеки во время PCR-FREE-получения индексированной библиотеки, а поэтому в нижеследующих стадиях типирования полученные данные секвенирования последовательно идентифицируют для каждого образца по индексам праймеров и индексам адаптеров, используемых в каждом образце. Последовательности каждого образца выравнивают с известной эталонной последовательностью ДНК, соответствующей ПЦР-продукту, с помощью компьютерной программы. Исходя из перекрывания последовательностей и характера их сцепления, сборку интактной последовательности для ПЦР-продукта осуществляют из последовательностей фрагментированной ДНК.
Настоящее изобретение относится к методам HLA-генотипирования высокого разрешения, осуществляемым путем секвенирования на Illumina GA, и, тем самым, к секвенированию гаплотипов, а также к автоматизации компьютерной программы типирования, к повышению производительности HLA-генотипирования и к снижению их стоимости.
Из-за требований, предъявляемых к длине ДНК-матрицы в современных методах секвенирования, и из-за небольшой длины ридов, исходные ПЦР-праймеры, используемые в методах HLA-SBT, больше не могут быть применены в HLA-типировании высокого разрешения, основанном на новых методах секвенирования. В настоящем изобретении были сконструированы новые ПЦР-праймеры с хорошей специфичностью и консервативностью, которые позволяют независимо амплифицировать экзоны 2, 3, 4 гена HLA-A, B и дают ПЦР-продукты с длиной не более чем 700 п.н., которые могут быть, в частности, использованы в секвенаторе Illumina GA (максимальная длина ДНК, подходящая для современного секвенатора Illumina GA, составляет 700 п.н.). Настоящее изобретение относится к набору ПЦР-праймеров, подходящих для широкомасштабного, высокопроизводительного и не дорогостоящего HLA-генотипирования у индивидуумов (в частности, у человека).
В техническом решении, используемом в настоящем изобретении, все наиболее поздние последовательности гена HLA-A/B загружают из web-сайта Интернета IMGT/HLA (http://www.ebi.ac.uk/imgt/hla/), а затем сохраняют на локальном диске как набор данных HLA-A; при этом все самые последние последовательности гена HLA-I, отличающиеся от последовательностей HLA-A, загружают как набор данных для сравнения. Указанные два набора данных сравнивают для поиска консервативных и специфических последовательностей для каждого генного сайта в двух концевых и внутренних частях экзонов 2, 3, 4, и сконструированную последовательность ПЦР-праймера сравнивают с полноразмерной последовательностью человеческого генома для поиска гомологии. Поскольку последовательность гена HLA-A/B имеет высокую степень сходства с последовательностями других генов, принадлежащих к молекулам HLA-I, то при конструировании ПЦР-праймеров 3'-конец праймера должен иметь, если это возможно, такую специфичность, которая гарантировала бы специфичность амплификации гена HLA-A/B с использованием этих праймеров. При этом длина ПЦР-продуктов составляет менее чем 700 п.н., а температура отжига прямого и обратного праймеров, по существу, является одинаковой.
Множество пар-кандидатов на праймеры HLA-A/B, удовлетворяющие требованиям протокола, используют для амплификации матричных ДНК, принадлежащих к общим серотипам HLA-A/B. Из этих праймеров было скринировано два набора ПЦР-праймеров HLA-A/B (по 6 пар для каждого набора) с наибольшей консервативностью и специфичностью, для их применения в целях амплификации экзонов 2, 3 и 4, соответственно.
Два набора ПЦР-праймеров (по 6 пар для каждого набора) были использованы в качестве базовых праймеров, на основе которых было сконструировано 95 наборов индексированных праймеров, используемых для амплификации 95 и 950 ДНК-матриц общих серотипов HLA-A/B (серотипами этих матриц являются все общие серотипы HLA-A/B), соответственно. Все ПЦР-продукты секвенировали по обоим концам в секвенаторе Illumina GA Pair-End 100, а затем смешивали в равных количествах, и после сборки, результаты секвенирования сравнивали с первоначальными результатами типирования для подтверждения консервативности и специфичности ПЦР-праймеров.
Праймеры HLA-A, B, сконструированные в соответствии с настоящим изобретением, то есть два набора ПЦР-праймеров HLA-A/B (по 6 пар для каждого набора) для амплификации экзонов 2, 3 и 4, соответственно, представлены в таблицах 1 и 2.
ПЦР-праймеры HLA-A,B
No.
ПЦР-праймеры HLA-A,B
Вырожденность праймеров означает смесь всех возможных различных последовательностей, представляющих собой все различные основания, кодирующие одну аминокислоту. Для повышения специфичности вырожденность может быть снижена путем замены оснований, встречающихся в различных организмах, основаниями в соответствии с таблицей кодонов, где R=A/G, Y=C/T, M=A/C, K=G/T, S=C/G, W=A/T, H=A/C/T, B=C/G/T, V=A/C/G, D=A/G/T, N=A/C/G/T.
В настоящем изобретении были сконструированы 2 набора ПЦР-праймеров (по три пары для каждого набора) для амплификации экзонов 2, 3 и 4 HLA-C с применением метода конструирования ПЦР-праймеров для амплификации экзонов 2, 3 и 4 гена HLA-A/B.
В нижеследующих примерах 95 и 950 проб крови с известными HLA-генотипами подвергали ПЦР-амплификации генов HLA-C с использованием 2 отобранных наборов ПЦР-праймеров (по 3 пары для каждого набора), соответственно. Амплифицированные продукты секвенировали методом Сэнгера и методом секвенирования второго поколения. Результаты секвенирования использовали для HLA-C-типирования и сравнивали с исходными результатами типирования для подтверждения консервативности и специфичности ПЦР-праймеров.
Настоящее изобретение относится к 2 наборам ПЦР-праймеров (по три пары для каждого набора) для амплификации экзонов 2, 3 и 4 гена HLA-C, которые представляют собой SEQ ID NO: 25 и 26, 27 и 28, и 29 и 30, как показано в таблице 3, и SEQ ID NO: 31 и 32, 33 и 34, и 35 и 36, как показано в таблице 4. Указанные 6 пар ПЦР-праймеров имеют хорошую консервативность и специфичность, и могут охватывать полноразмерные последовательности экзонов 2, 3 и 4 HLA-C, где длина всех ПЦР-продуктов составляет менее чем 700 п.н., что удовлетворяет требованиям стандартного метода секвенирования Illumina Solexa. Кроме того, праймеры согласно изобретению могут быть также использованы для секвенирования методом Сэнгера.
ПЦР-праймеры экзонов 2, 3 и 4 гена HLA-C
ПЦР-праймеры экзонов 2, 3 и 4 гена HLA-C
В соответствии с описанными выше методами, применяемыми на секвенаторе второго поколения для генотипирования HLA-DQB1, настоящее изобретение относится к ПЦР-праймерам для амплификации экзонов 2 и/или 3 HLA-DQB1, которые имеют последовательности SEQ ID NO: 37-40, представленные в таблице 5. Эти ПЦР-праймеры имеют хорошую консервативность и специфичность, и могут охватывать полноразмерные последовательности экзонов 2, 3 и 4 HLA-DQB1, где длина всех ПЦР-продуктов составляет менее чем 700 п.н., что удовлетворяет требованиям стандартного метода секвенирования Illumina Solexa. Кроме того, праймеры согласно изобретению могут быть также использованы для секвенирования по Сэнгеру.
ПЦР-праймеры для амплификации соответствующих экзонов HLA-DQB1
Генотипирование может быть осуществлено путем амплификации экзонов 2 и/или 3 HLA-DQB1 с применением пары праймеров для амплификации, и настоящее изобретение относится к таким методам генотипирования. По сравнению с ранее применяемыми методами, в таком генотипировании используется технология Illumina Solexa, которая позволяет проводить высокоразрешающее HLA-типирование с высокой производительностью и низкими затратами.
Конкретные способы осуществления изобретения
Способ секвенирования нуклеиновой кислоты
В одном из своих аспектов настоящее изобретение относится к способу определения нуклеотидной последовательности представляющей интерес нуклеиновой кислоты в образце, где указанный способ включает:
1) получение n образцов, где n представляет собой целое число ≥1, а указанные образцы предпочтительно берут у млекопитающего, более предпочтительно у человека, в частности, такими образцами являются пробы человеческой крови; причем необязательно указанные n образцов, предназначенные для анализа, разделяют на m групп, где m представляет собой целое число, а n≥m≥1;
2) амплификацию, где пару или множество пар индексированных праймеров используют для каждого образца, если от этого образца имеются матрицы; ПЦР-амплификацию осуществляют в условиях, подходящих для амплификации представляющей интерес нуклеиновой кислоты, где каждая пара индексированных праймеров состоит из прямого индексированного праймера и обратного индексированного праймера (оба эти праймера могут быть вырожденными), содержащих праймерные индексы, где указанные праймерные индексы, присутствующие в прямом индексированном праймере и в обратном индексированном праймере, могут быть одинаковыми или различными, то есть индексы праймеров в парах индексированных праймеров, используемых для различных образцов, являются различными;
3) объединение в пул: если n>1, то ПЦР-продукты от каждого образца объединяют в один пул;
4) фрагментирование, где амплифицированные продукты подвергают неполному фрагментированию, очистке и выделению;
5) секвенирование, где выделенную ДНК-смесь подвергают секвенированию с применением технологии секвенирования второго поколения, предпочтительно парно-концевым методом (например, Illumina GA, Illumina Hiseq 2000), с получением последовательностей фрагментированной ДНК; и
6) сборку, где данные секвенирования, полученные для образцов, последовательно сопоставляют по уникальному праймерному индексу для каждого образца; каждую считанную последовательность сопоставляют путем выравнивания с эталонной последовательностью ДНК, соответствующей ПЦР-продуктам, с помощью программы выравнивания (такой как программа Blast, BWA); сборку полноразмерной последовательности представляющей интерес нуклеиновой кислоты осуществляют из последовательностей фрагментированной ДНК по перекрыванию и характеру сцепления последовательностей.
В одном из аспектов настоящего изобретения каждая пара праймерных индексов и пара ПЦР-праймеров образует пару индексированных праймеров, где прямой и обратный ПЦР-праймеры имеют индекс прямого праймера и индекс обратного праймера у 5'-конца (или, необязательно, присоединенный посредством линкерной последовательности), соответственно.
В одном из вариантов изобретения указанными ПЦР-праймерами являются ПЦР-праймеры для амплификации гена HLA, в частности ПЦР-праймеры для амплификации гена HLA-A/B, предпочтительно ПЦР-праймеры для амплификации экзонов 2, 3 и 4 HLA-A/B и экзона 2 HLA-DRB1, предпочтительно ПЦР-праймеры для амплификации экзонов 2, 3 и 4 HLA-A/B, как показано в таблице 1 или в таблице 2, или предпочтительно ПЦР-праймеры для амплификации экзона 2 HLA-DRB1, как показано в таблице 7.
В одном из вариантов изобретения указанными ПЦР-праймерами являются ПЦР-праймеры для амплификации гена HLA, в частности ПЦР-праймеры для амплификации гена HLA-С, предпочтительно ПЦР-праймеры для амплификации экзонов 2, 3 и 4 HLA-С, где предпочтительные указанные ПЦР-праймеры представлены в таблице 3 или в таблице 4.
В одном из вариантов изобретения указанными ПЦР-праймерами являются ПЦР-праймеры для амплификации гена HLA, предпочтительно ПЦР-праймеры для амплификации экзона 2 и/или 3 гена HLA-DQB1; где предпочтительные указанные ПЦР-праймеры представлены в таблице 5.
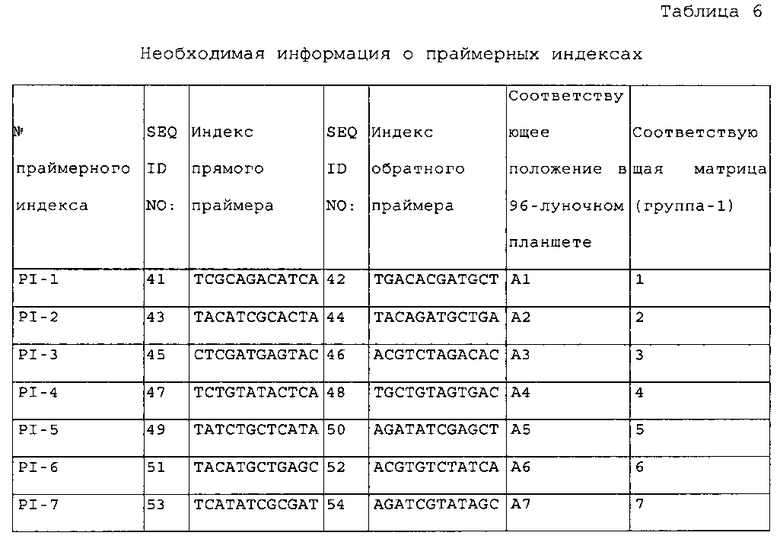
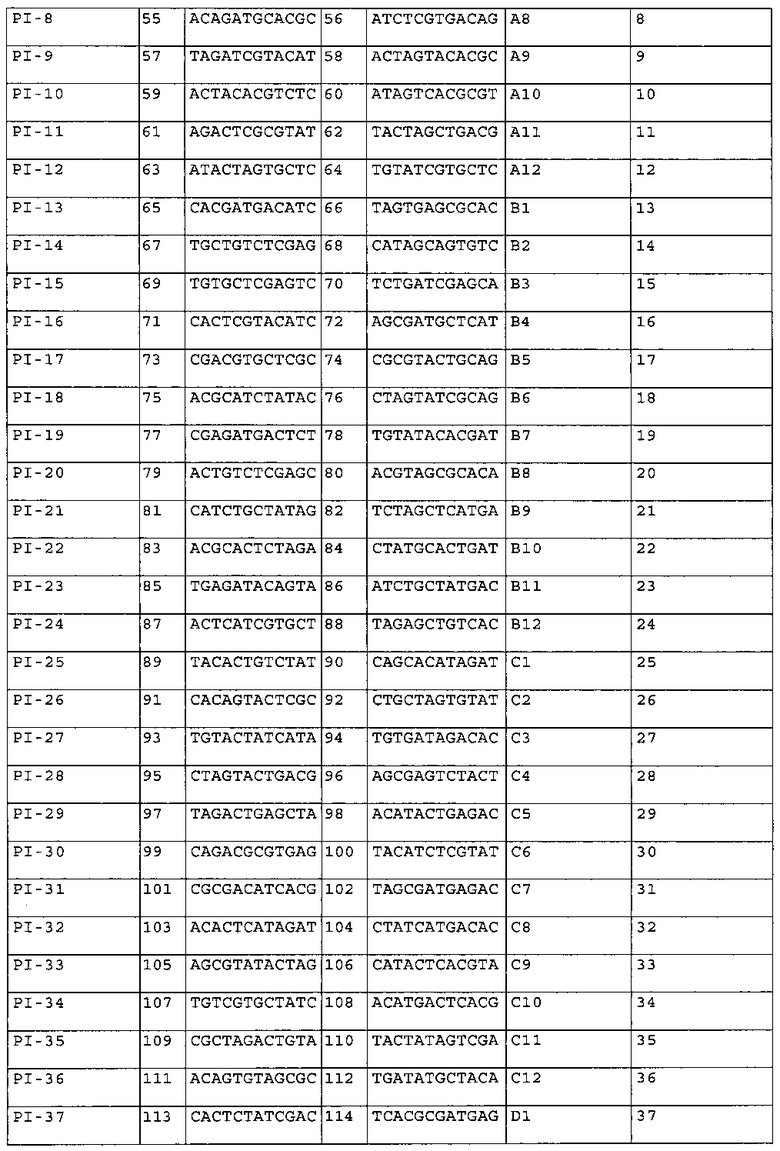
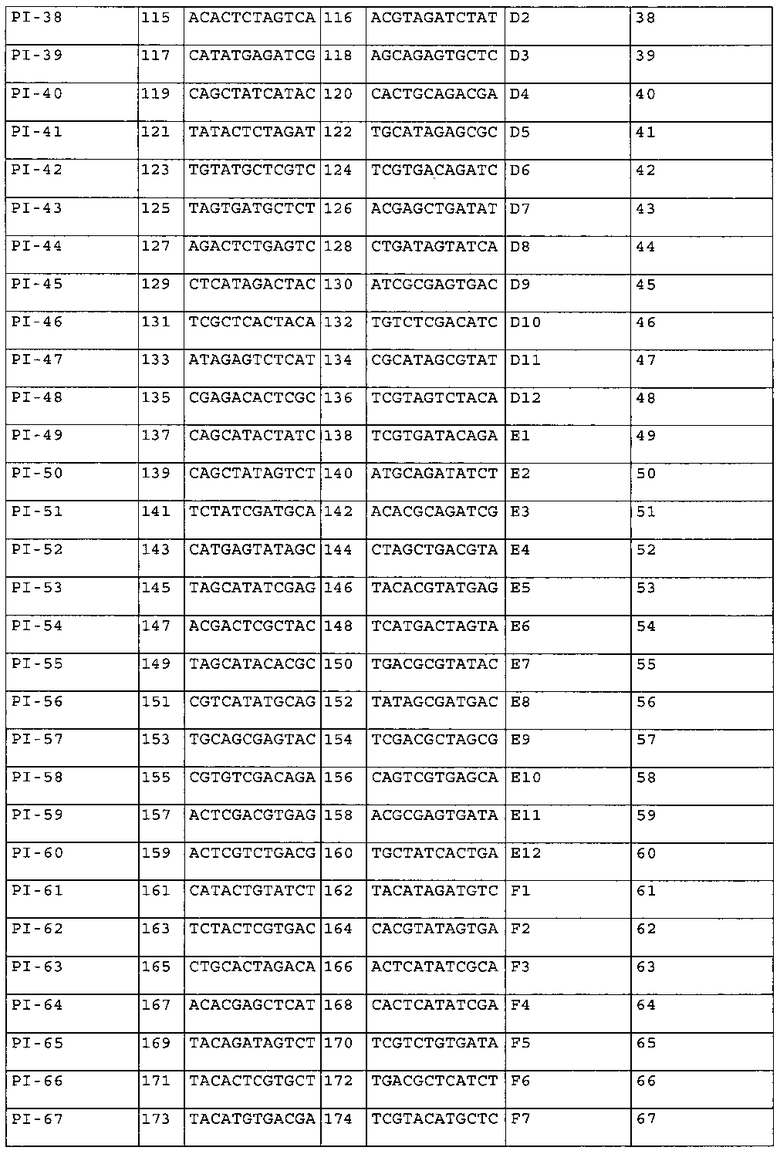
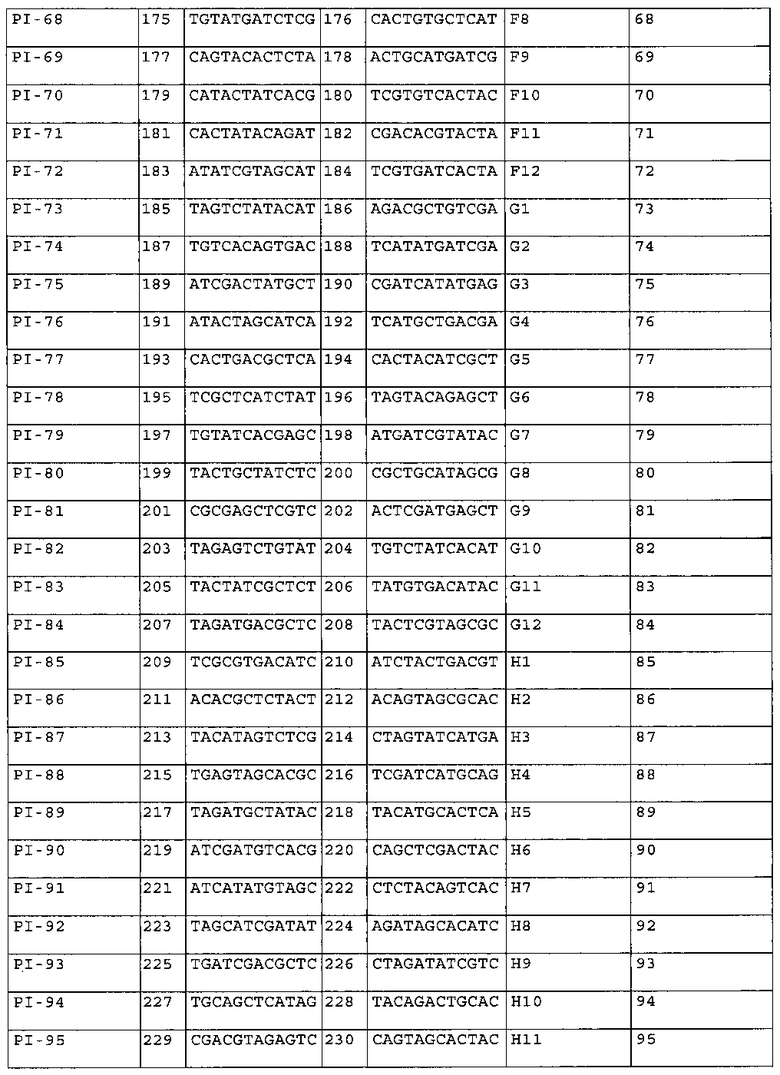
В одном из аспектов изобретения указанные праймерные индексы были сконструированы для ПЦР-праймеров, предпочтительно для ПЦР-праймеров, используемых для амплификации специфического гена HLA, более предпочтительно для ПЦР-праймеров, используемых для амплификации экзонов 2, 3 и 4 HLA-A/B и экзона 2 для HLA-DRB1, в частности, для ПЦР-праймеров, представленных в таблице 1, в таблице 2 или в таблице 7; где указанные праймерные индексы, в частности, содержат по меньшей мере 10, или по меньшей мере 20, или по меньшей мере 30, или по меньшей мере 40, или по меньшей мере 50, или по меньшей мере 60, или по меньшей мере 70, или по меньшей мере 80, или по меньшей мере 90, или все 95 пар из 95 пар праймерных индексов, представленных в таблице 6 (или набор праймерных индексов, состоящий из 10-95 пар (например, 10-95 пар, 20-95 пар, 30-95 пар, 40-95 пар, 50-95 пар, 60-95 пар, 70-95 пар, 80-95 пар, 90-95 пар, или 95 пар) из 95 пар праймерных индексов, представленных в таблице 6); и
набор индексированных праймеров предпочтительно включает по меньшей мере PI-1 - PI-10, или PI-11 - PI-20, или PI-21 - PI-30, или PI-31 - PI-40, или PI-41 - PI-50, или PI-51 - PI-60, или PI-61 - PI-70, или PI-71 - PI-80, или PI-81 - PI-90, или PI-91 - PI-95 из 95 пар праймерных индексов, представленных в таблице 6, или комбинации любых двух или более из указанных пар.
В одном из вариантов изобретения указанное фрагментирование ДНК включает методы химического фрагментирования и методы физического фрагментирования, где методами химического фрагментирования является ферментативное расщепление, а методами физического фрагментирования являются методы ультразвукового фрагментирования или методы механического фрагментирования.
В одном из вариантов изобретения, после указанного фрагментирования ДНК, все ДНК-полосы, имеющие длину между максимальной длиной ридов в секвенаторе и максимально приемлемой длиной ДНК в секвенаторе, очищают и выделяют, где указанными методами очистки и выделения являются, но не ограничиваются ими, выделение путем электрофореза и разрезания геля, и выделение на магнитных сферах.
В другом варианте изобретения способ секвенирования нуклеотидной последовательности представляющей интерес нуклеиновой кислоты в тест-образце включает стадии 1)-4) по п.1 формулы изобретения, и нижеследующие стадии:
5) конструирование библиотеки, а именно, конструирование PCR-FREE-библиотеки для секвенирования с использованием библиотеки фрагментированных ПЦР-продуктов, где различные адаптеры библиотеки могут быть добавлены для распознавания различных PCR-FREE-библиотек для секвенирования, и где все ДНК-полосы, имеющие длину между максимальной длиной ридов в секвенаторе и максимально приемлемой длиной ДНК в секвенаторе, предпочтительно ДНК-фрагменты длиной 450-750 п.н., очищают и выделяют;
6) секвенирование, где выделенную ДНК-смесь подвергают секвенированию с применением технологии секвенирования второго поколения, предпочтительно технологии парно-концевого секвенирования (например, на секвенаторе Illumina GA, Illumina Hiseq 2000), с получением последовательностей фрагментированной ДНК; и
7) сборка, где данные секвенирования, полученные для образцов, последовательно сопоставляют по различным последовательностям адаптеров библиотеки и по уникальному праймерному индексу для каждого образца; каждую считанную последовательность сопоставляют путем выравнивания с эталонной последовательностью ДНК, соответствующей ПЦР-продуктам, с помощью программы выравнивания (такой как программа Blast, BWA); и сборку полноразмерной последовательности представляющей интерес нуклеиновой кислоты осуществляют из последовательностей фрагментированной ДНК по перекрыванию и характеру сцепления последовательностей.
В одном из своих аспектов настоящее изобретение также относится к применению вышеописанного способа для HLA-типирования, где указанный способ отличается тем, что он включает: секвенирование взятого у пациента образца (в частности, пробы крови) указанным методом и сопоставление результатов секвенирования с данными о последовательностях экзонов HLA, предпочтительно экзонов 2, 3, 4 HLA-A/B, экзонов 2, 3 и/или 4 HLA-C, экзона 2 и/или 3 гена HLA-DQB1 и/или экзона 2 HLA-DRB1 в базе данных HLA (такой как профессиональная база данных HLA IMGT); где если результат выравнивания последовательностей дает 100% соответствие, то это означает, что HLA-генотип соответствующего образца был точно определен.
Набор праймерных индексов
В другом своем аспекте настоящее изобретение относится к набору праймерных индексов, где указанный набор содержит по меньшей мере 10, или по меньшей мере 20, или по меньшей мере 30, или по меньшей мере 40, или по меньшей мере 50, или по меньшей мере 60, или по меньшей мере 70, или по меньшей мере 80, или по меньшей мере 90, или все 95 пар из 95 пар праймерных индексов, представленных в таблице 6 (или набор праймерных индексов, состоящий из 10-95 пар (например, 10-95 пар, 20-95 пар, 30-95 пар, 40-95 пар, 50-95 пар, 60-95 пар, 70-95 пар, 80-95 пар, 90-95 пар, или 95 пар) из 95 пар праймерных индексов, представленных в таблице 6); и
указанный набор индексированных праймеров предпочтительно включает по меньшей мере PI-1 - PI-10, или PI-11 - PI-20, или PI-21 - PI-30, или PI-31 - PI-40, или PI-41 - PI-50, или PI-51 - PI-60, или PI-61 - PI-70, или PI-71 - PI-80, или PI-81 - PI-90, или PI-91 - PI-95 из 95 пар праймерных индексов, представленных в таблице 6, или комбинации любых двух или более из указанных пар.
Настоящее изобретение также относится к применению указанного набора праймерных индексов в методах ПЦР-секвенирования, где, в частности, каждая пара праймерных индексов и пара ПЦР-праймеров для амплификации представляющей интерес тестируемой последовательности образует пару индексированных праймеров, где прямой и обратный ПЦР-праймеры имеют индекс прямого праймера или индекс обратного праймера у 5'-конца (или, необязательно, присоединенный посредством линкерной последовательности), соответственно.
В одном из аспектов изобретения указанными ПЦР-праймерами являются ПЦР-праймеры для амплификации специфического гена HLA, в частности, ПЦР-праймеры для амплификации экзонов 2, 3 и 4 HLA-A/B и экзона 2 HLA-DRB1, предпочтительно ПЦР-праймеры для амплификации экзонов 2, 3 и 4 HLA-A/B, как показано в таблице 1 или в таблице 2, или предпочтительно ПЦР-праймеры для амплификации экзона 2 HLA-DRB1, как показано в таблице 7, или предпочтительно ПЦР-праймеры для амплификации экзонов 2, 3 и/или 4 HLA-С, где предпочтительные указанные ПЦР-праймеры представлены в таблице 3 или в таблице 4, или предпочтительно ПЦР-праймеры для амплификации экзона 2 и/или 3 гена HLA-DQB1, где предпочтительные указанные ПЦР-праймеры представлены в таблице 5.
В другом своем аспекте настоящее изобретение относится к набору индексированных праймеров, содержащему указанный набор праймерных индексов и пару ПЦР-праймеров для амплификации представляющей интерес тестируемой последовательности, где пара индексированных праймеров содержит пару праймерных индексов и пару ПЦР-праймеров, где прямой и обратный ПЦР-праймеры имеют индекс прямого праймера и индекс обратного праймера у 5'-конца (или, необязательно, присоединенный посредством линкерной последовательности), соответственно.
В одном из вариантов изобретения, указанными ПЦР-праймерами являются ПЦР-праймеры для амплификации специфического гена HLA, в частности, ПЦР-праймеры для амплификации экзонов 2, 3 и 4 гена HLA-A/B и экзона 2 HLA-DRB1, предпочтительно ПЦР-праймеры для амплификации экзонов 2, 3 и 4 HLA-A/B, как показано в таблице 1 или в таблице 2, или предпочтительно ПЦР-праймеры для амплификации экзона 2 HLA-DRB1, как показано в таблице 7, предпочтительно ПЦР-праймеры для амплификации экзонов 2, 3 и/или 4 HLA-С, где предпочтительные указанные ПЦР-праймеры представлены в таблице 3 или в таблице 4, или предпочтительно ПЦР-праймеры для амплификации экзона 2 и/или 3 гена HLA-DQB1, где предпочтительные указанные ПЦР-праймеры представлены в таблице 5.
В другом своем аспекте настоящее изобретение также относится к применению указанных индексированных праймеров в методах ПЦР-секвенирования.
Способ HLA-типирования
В одном из своих аспектов настоящее изобретение относится к способу HLA-типирования, включающему:
1) получение n образцов, где n представляет собой целое число ≥1, а указанные образцы предпочтительно, берут у млекопитающего, более предпочтительно у человека, в частности, такими образцами являются пробы человеческой крови;
2) разделение анализируемых n образцов на m групп, где m равно целому числу, а n≥m≥1;
3) амплификацию, где пару индексированных праймеров используют для каждого образца, если имеются матрицы от этого образца; ПЦР-амплификацию осуществляют в условиях, подходящих для амплификации представляющей интерес нуклеиновой кислоты, где каждая пара индексированных праймеров состоит из прямого индексированного праймера и обратного индексированного праймера (оба эти праймера могут быть вырожденными), содержащих праймерные индексы, где праймерные индексы, присутствующие в прямом индексированном праймере и в обратном индексированном праймере, могут быть одинаковыми или различными, то есть индексы праймеров в парах индексированных праймеров, используемых для различных образцов, являются различными;
4) объединение в пул, где продукты ПЦР-амплификации от каждого образца объединяют в один пул с получением библиотек ПЦР-продуктов;
5) фрагментирование, где полученные библиотеки ПЦР-продуктов подвергают неполному фрагментированию;
6) конструирование библиотек, а именно, конструирование PCR-FREE-библиотек для секвенирования из библиотеки фрагментированных ПЦР-продуктов с применением технологии индексирования адаптеров библиотек, где различные адаптеры библиотеки могут быть добавлены для распознавания различных PCR-FREE-библиотек для секвенирования, и все ДНК-полосы, имеющие длину между максимальной длиной ридов в секвенаторе и максимально приемлемой длиной ДНК в секвенаторе, предпочтительно ДНК-фрагменты длиной 450-750 п.н., выделяют;
7) секвенирование, где выделенную ДНК-смесь подвергают секвенированию с применением технологии секвенирования второго поколения, предпочтительно технологии парно-концевого секвенирования (например, на Illumina GA, Illumina Hiseq 2000), с получением последовательностей фрагментированных ДНК; и
8) сборку, где данные секвенирования, полученные для образцов, последовательно сопоставляют по различным последовательностям адаптеров библиотеки и по уникальному праймерному индексу для каждого образца; каждый рид сопоставляют путем выравнивания с эталонной последовательностью ДНК, соответствующей ПЦР-продуктам, с помощью программы выравнивания (такой как программа Blast, BWA); сборку полноразмерной последовательности представляющей интерес нуклеиновой кислоты осуществляют из последовательностей фрагментированной ДНК по перекрыванию и характеру сцепления последовательностей; и
9) типирование, где результаты секвенирования сопоставляют с данными о последовательностях экзонов HLA, предпочтительно экзонов 2, 3, 4 HLA-A/B, экзонов 2, 3 и/или 4 HLA-C, экзона 2 и/или 3 гена HLA-DQB1 и/или экзона 2 HLA-DRB1 в базе данных HLA (такой как профессиональная база данных HLA IMGT); при этом если результат выравнивания последовательностей дает 100% соответствие, то это означает, что HLA-генотип соответствующего образца был точно определен.
В способе HLA-типирования согласно изобретению, пара индексированных праймеров содержит пару праймерных индексов и пару ПЦР-праймеров, где прямой и обратный ПЦР-праймеры имеют индекс прямого праймера и индекс обратного праймера у 5'-конца (или, необязательно, присоединены посредством линкерной последовательности), соответственно.
В одном из вариантов изобретения указанными ПЦР-праймерами являются ПЦР-праймеры для амплификации специфического гена HLA, в частности, ПЦР-праймеры для амплификации экзонов 2, 3 и 4 гена HLA-A/B и экзона 2 HLA-DRB1, предпочтительно ПЦР-праймеры для амплификации экзонов 2, 3 и 4 HLA-A/B, как показано в таблице 1 или в таблице 2, или предпочтительно ПЦР-праймеры для амплификации экзона 2 HLA-DRB1, как показано в таблице 7, или предпочтительно ПЦР-праймеры для амплификации экзонов 2, 3 и/или 4 HLA-С, где предпочтительные указанные ПЦР-праймеры представлены в таблице 3 или в таблице 4, или предпочтительно ПЦР-праймеры для амплификации экзона 2 и/или 3 гена HLA-DQB1; где предпочтительные указанные ПЦР-праймеры представлены в таблице 5.
В одном из вариантов изобретения указанные праймерные индексы представляют собой набор праймерных индексов, описанных выше.
В одном из вариантов способа HLA-типирования согласно изобретению, указанное фрагментирование ДНК включает методы химического фрагментирования и методы физического фрагментирования, где методами химического фрагментирования является ферментативное расщепление, а методами физического фрагментирования являются методы ультразвукового фрагментирования или методы механического фрагментирования.
В одном из вариантов способа HLA-типирования согласно изобретению, указанными методами очистки и выделения являются, но не ограничиваются ими, выделение путем электрофореза и разрезания геля, и выделение на магнитных сферах.
В одном из вариантов способа HLA-типирования согласно изобретению, конструирование библиотек для PCR-FREE-секвенирования из библиотек фрагментированных ПЦР-продуктов методом индексирования адаптера библиотеки включает: добавление m адаптеров библиотеки к m библиотекам ПЦР-продуктов, полученных в стадии 2), где в каждой библиотеке ПЦР-продуктов используется другой адаптер библиотеки; и конструирование m библиотек индексированных адаптеров для секвенирования; объединение в пул m библиотек индексированных адаптеров для секвенирования в равном молярном соотношении с получением смеси библиотек индексированных адаптеров для секвенирования, где указанный способ присоединения адаптеров библиотек означает прямое связывание посредством ДНК-лигазы без проведения ПЦР.
ПЦР-праймеры для HLA-генотипирования
В одном из своих аспектов настоящее изобретение относится к ПЦР-праймерам для HLA-генотипирования, где указанные праймеры отличаются тем, что они представляют собой ПЦР-праймеры для амплификации экзонов 2, 3 и 4 гена HLA-A/B и экзона 2 HLA-DRB1, предпочтительно ПЦР-праймеры для амплификации экзонов 2, 3 и 4 HLA-A/B, как показано в таблице 1 или в таблице 2, или предпочтительно ПЦР-праймеры для амплификации экзона 2 HLA-DRB1, как показано в таблице 7, предпочтительно ПЦР-праймеры для амплификации экзонов 2, 3 и/или 4 HLA-С, где предпочтительные указанные ПЦР-праймеры представлены в таблице 3 или в таблице 4, или предпочтительно ПЦР-праймеры для амплификации экзона 2 и/или 3 гена HLA-DQB1, где предпочтительные указанные ПЦР-праймеры представлены в таблице 5.
Настоящее изобретение также относится к способу секвенирования с использованием указанных ПЦР-праймеров, включающему:
получение образца, в частности, пробы крови, где указанную пробу крови берут предпочтительно у млекопитающего, в частности, у человека;
амплификацию, где амплификацию ДНК из пробы крови осуществляют с использованием ПЦР-праймеров с получением ПЦР-продуктов и очистку этих ПЦР-продуктов;
секвенирование, где ПЦР-продукты подвергают секвенированию, и где методом секвенирования может быть метод секвенирования по Сэнгеру или метод секвенирования второго поколения (такой как метод Hiseq 2000, Illumina GA и Roche454).
В другом своем аспекте настоящее изобретение также относится к применению указанных ПЦР-праймеров для HLA-генотипирования, отличающемуся тем, что оно включает: использование указанных ПЦР-праймеров, сборку и анализ путем сопоставления результатов, полученных вышеуказанным методом секвенирования; и сравнение результатов секвенирования со стандартными последовательностями, имеющимися в базе данных с получением результатов HLA-генотипирования.
В другом своем аспекте настоящее изобретение также относится к набору для HLA-генотипирования, содержащему указанные ПЦР-праймеры.
ПЦР-праймеры для HLA-A,B-генотипирования
В одном из своих аспектов настоящее изобретение относится к набору ПЦР-праймеров для HLA-A,B-генотипирования, где указанный набор отличается тем, что он содержит указанные ПЦР-праймеры, представленные в таблице 1 или в таблице 2.
В другом своем аспекте настоящее изобретение относится к способу секвенирования с использованием указанных ПЦР-праймеров для HLA-A,B-генотипирования, где указанный способ включает:
получение образца, в частности, пробы крови, где указанную пробу крови берут предпочтительно от млекопитающего, в частности, у человека;
амплификацию, где амплификацию ДНК из пробы крови осуществляют с использованием ПЦР-праймеров с получением ПЦР-продуктов и очистку этих ПЦР-продуктов;
секвенирование, где ПЦР-продукты подвергают секвенированию, и где методом секвенирования может быть метод секвенирования по Сэнгеру или метод секвенирования второго поколения (такой как метод Hiseq 2000, Illumina GA и Roche454).
В другом своем аспекте, настоящее изобретение также относится к применению указанных ПЦР-праймеров для HLA-генотипирования, отличающемуся тем, что оно включает использование указанных ПЦР-праймеров, сборку и анализ путем сопоставления результатов, полученных вышеуказанным методом секвенирования, и сравнение результатов секвенирования со стандартными последовательностями, имеющимися в базе данных, с получением результатов HLA-генотипирования.
В другом своем аспекте настоящее изобретение также относится к набору для HLA-генотипирования, содержащему указанные ПЦР-праймеры для HLA-A,B-генотипирования согласно изобретению.
ПЦР-праймеры для HLA-С-генотипирования
Настоящее изобретение также относится к новому способу амплификации экзонов 2, 3 и 4 гена HLA-C, отличающемуся тем, что он включает ПЦР-амплификацию с использованием пар праймеров для амплификации согласно изобретению, последовательности которых представлены в таблице 3 или в таблице 4.
Поскольку экзоны 2, 3 и 4 HLA-C могут быть амплифицированы с помощью ПЦР, то способ согласно изобретению является особенно подходящим для HLA-C-генотипирования. Поскольку указанные продукты были получены с применением такого способа, а праймеры для амплификации согласно изобретению имеют размеры в пределах 700 п.н., то в процессе последующего генотипирования может быть использован не известный уже метод HLA-C-генотипирования, а метод HLA-SBT, разработанный на основе технологии секвенирования Illumina Solexa.
Настоящее изобретение также относится к способу секвенирования экзонов 2, 3 и 4 гена HLA-C в образцах, где указанный способ включает следующие стадии:
1) получение образца и экстракция ДНК из этого образца;
2) амплификация ДНК с использованием пары ПЦР-праймеров для HLA-C-генотипирования согласно изобретению с получением ПЦР-продуктов, и предпочтительно очистка ПЦР-продуктов, где указанная пара ПЦР-праймеров предпочтительно выбрана из группы, состоящей из пары праймеров SEQ ID NO: 25 и SEQ ID NO: 26, SEQ ID NO: 27 и SEQ ID NO: 28, SEQ ID NO: 29 и SEQ ID NO: 30, или SEQ ID NO: 31 и SEQ ID NO: 32, SEQ ID NO: 33 и SEQ ID NO: 34, SEQ ID NO: 35 и SEQ ID NO: 36;
3) секвенирование ПЦР-продуктов предпочтительно методом секвенирования второго поколения, таким как Illumina Solexa или Roche454.
Настоящее изобретение также относится к способу HLA-C-генотипирования, включающему:
1) ПЦР-амплификацию экзонов 2, 3 и/или 4 гена HLA-C тестируемого образца с использованием пары ПЦР-праймеров для HLA-C-генотипирования согласно изобретению, где указанная пара ПЦР-праймеров предпочтительно выбрана из группы, состоящей из пар праймеров SEQ ID NO: 25 и SEQ ID NO: 26, SEQ ID NO: 27 и SEQ ID NO: 28, SEQ ID NO: 29 и SEQ ID NO: 30, или SEQ ID NO: 31 и SEQ ID NO: 32, SEQ ID NO: 33 и SEQ ID NO: 34, SEQ ID NO: 35 и SEQ ID NO: 36;
2) секвенирование амплифицированных экзонов; сравнение результатов секвенирования с результатами для стандартных последовательностей, имеющихся в базе данных, и получение результатов генотипирования, где указанное секвенирование осуществляют методом секвенирования по Сэнгеру или методом секвенирования второго поколения, таким как Illumina Solexa или Roche454.
В другом своем аспекте настоящее изобретение также относится к набору для HLA-C-генотипирования, включающему пару ПЦР-праймеров для HLA-С-генотипирования согласно изобретению, предпочтительно выбранную из группы, состоящей из пар праймеров: SEQ ID NO: 25 и SEQ ID NO: 26, SEQ ID NO: 27 и SEQ ID NO: 28, SEQ ID NO: 29 и SEQ ID NO: 30, или SEQ ID NO: 31 и SEQ ID NO: 32, SEQ ID NO: 33 и SEQ ID NO: 34, SEQ ID NO: 35 и SEQ ID NO: 36. В одном из вариантов изобретения указанный набор также включает дополнительные агенты, например, агенты для амплификации ДНК, очистки ДНК и/или секвенирования ДНК.
Генотипирование может быть осуществлено посредством амплификации экзонов 2, 3 и 4 HLA-C с использованием пары праймеров для амплификации и с применением способа генотипирования согласно изобретению. Способ генотипирования согласно изобретению, по сравнению с известным методом, может быть проведен с применением метода секвенирования Illumina Solexa, способствует повышению производительности, упрощает процедуру и при этом позволяет экономить время и средства.
ПЦР-праймеры для HLA-DQB1-генотипирования
Настоящее изобретение также относится к новому способу амплификации экзона 2 и/или 3 HLA-DQB1, отличающемуся тем, что он включает ПЦР-амплификацию с использованием пар праймеров для амплификации согласно изобретению, где указанные пары праймеров для амплификации представлены в таблице 5.
Поскольку экзоны 2 и/или 3 HLA-DQB1 могут быть амплифицированы с помощью ПЦР, то способ согласно изобретению является особенно подходящим для HLA-DQB1-генотипирования. Поскольку указанные продукты были получены с применением такого способа, и праймеры для амплификации согласно изобретению имеют размеры в пределах 300-400 п.н., то в процессе последующего типирования может быть применен не известный метод HLA-DQB1-генотипирования, а метод HLA-SBT на основе технологии секвенирования Illumina Solexa.
Настоящее изобретение также относится к способу секвенирования экзонов 2 и/или 3 гена HLA-DQB1 в образцах, включающему нижеследующие стадии:
1) получение образца и экстракция ДНК из этого образца;
2) амплификация ДНК с использованием пары ПЦР-праймеров для HLA-DQB1-генотипирования согласно изобретению, предпочтительно с использованием пар ПЦР-праймеров, представленных в таблице 5, с получением ПЦР-продуктов, и предпочтительно очистка этих ПЦР-продуктов;
3) секвенирование ПЦР-продуктов, предпочтительно методом секвенирования второго поколения, таким как Illumina Solexa или Roche454.
В другом своем аспекте настоящее изобретение относится к усовершенствованному способу HLA-DQB1-генотипирования, включающему:
1) амплификацию тестируемых экзонов 2 и/или 3 гена HLA-DQB1 с использованием пары ПЦР-праймеров для HLA-DQB1-генотипирования согласно изобретению, предпочтительно с использованием пар ПЦР-праймеров, представленных в таблице 5;
2) секвенирование амплифицированных экзонов; сравнение результатов секвенирования со стандартными последовательностями, имеющимися в базе данных; и получение результатов генотипирования, где указанным способом секвенирования может быть метод секвенирования Сэнгера или метод секвенирования второго поколения, такой как Illumina Solexa или Roche454.
В другом своем аспекте настоящее изобретение также относится к набору для HLA-DQB1-генотипирования, включающему пару ПЦР-праймеров для HLA-DQB1-генотипирования согласно изобретению, предпочтительно пары ПЦР-праймеров для амплификации, представленные в таблице 5. В одном из вариантов изобретения указанный набор также включает дополнительные агенты, например, агенты для амплификации ДНК, очистки ДНК и/или секвенирования ДНК.
Описание графического материала
Фигура 1. На этой фигуре проиллюстрирована сборка последовательностей после мечения праймерными индексами, фрагментирования ДНК и секвенирования ДНК. Последовательности индексов прямого и обратного праймеров, Индекс-N-F/R (1), вводят в оба конца ПЦР-продуктов образца No. N. После фрагментирования физическим методом, ПЦР-продукты включают продукты, несущие последовательности праймерных индексов у одного конца, продукты, не содержащие последовательности праймерных индексов у своих двух концов, и полностью нефрагментированные продукты. Все ДНК-полосы, имеющие длину между максимальной длиной ридов секвенатора и максимально приемлемой длиной ДНК в секвенаторе, очищают и выделяют путем разрезания геля, а затем используют для секвенирования (2). Данные секвенирования ПЦР-продуктов, принадлежащих к образцу No. N, метят с использованием последовательности Индекс-N-F/R. Известные эталонные последовательности ПЦР-продуктов используют для определения относительной локализации положений считываемых последовательностей, и результаты секвенирования полноразмерных ПЦР-продуктов собирают с учетом перекрывания и характера сцепления считываемых последовательностей (3, 4).
Фигура 2. На этой фигуре проиллюстрированы результаты электрофореза ПЦР-продуктов соответствующих экзонов HLA-A/B/DRB1 в образце No.1 примера 2. На электрофореграмме видно, что ПЦР-продукты представляют собой серию отдельных полос размером 300 п.н. - 500 п.н., где дорожка M представляет собой маркер молекулярной массы (DL 2000, Takara Co.), дорожки 1-7 представляют собой ПЦР-продукты экзонов (A2, A3, A4, B2, B3, B4, DRB1-2) HLA-A/B/DRB1 образца No.1, а в негативном контроле (N) полоса амплификации отсутствует. Другие образцы показывают аналогичные результаты.
Фигура 3. На этой фигуре проиллюстрированы результаты электрофореза ДНК после фрагментирования HLA-Mix, как описано в примере 4 (до и после разрезания геля), где площадь гелевых срезов составляет 450-750 п.н. Дорожка M представляет собой маркер молекулярной массы (ДНК-лэддер NEB-50 п.н.), дорожка 1 представляет собой результаты электрофореза HLA-Mix до разрезания геля, а дорожка 2 представляет собой гель с HLA-Mix после разрезания.
Фигура 4. Программа с захватом изображения на экране для конструирования консенсусной последовательности образца No.1 примера 6, иллюстрирующая сборку полноразмерной последовательности ПЦР-продуктов на основе праймерных индексов и перекрывания ДНК-фрагментов. Номенклатуру HLA-генотипов можно найти на сайте http://www.ebi.ac.uk/imgt/hla/align.html. Результаты для всей кодирующей последовательности A*02:03:01 A*11:01:01 приводятся в столбце выходных данных слева, где последовательность экзона 2 идентична исходному известному результату для Матрицы 1.
Фигура 5. На этой фигуре проиллюстрирован ПЦР-продукт после мечения праймерными индексами и адаптерным индексом. Во время эксперимента праймерные индексы вводят в оба конца ПЦР-продукта каждого образца одновременно с помощью ПЦР, а затем множество ПЦР-продуктов, несущих различные праймерные индексы, объединяют в пул и конструируют библиотеку для секвенирования. В процессе конструирования библиотек для секвенирования, в том случае, если необходимо сконструировать множество таких библиотек, эти библиотеки могут быть помечены соответствующими адаптерами, несущими различные адаптерные индексы. После завершения конструирования библиотек, множество библиотек для секвенирования, меченных различными адаптерными индексами, объединяют в пул и одновременно секвенируют в секвенаторе Illumina GA (праймерные индексы могут быть идентичными в различных библиотеках для секвенирования, помеченных различными адаптерными индексами). После получения результатов секвенирования, информация о последовательностях ДНК для каждого образца может быть получена путем скрининга результатов секвенирования в целях поиска информации о последовательностях адаптерных индексов и праймерных индексов.
Фигура 6. На этой фигуре проиллюстрированы результаты электрофореза ПЦР-продуктов для экзонов 2, 3, 4 гена HLA-C некоторых образцов примера 8. На электрофореграмме видно, что ПЦР-продукты представляют собой серию отдельных полос размером 400-500 п.н., где дорожка M представляет собой эталон молекулярных масс стандартных ДНК (DL 2000, Takara Co.).
Фигура 7. На этой фигуре проиллюстрированы результаты электрофореза ДНК с разрезанием геля после фрагментирования HLA-Mix, как описано в примере 8, где площадь гелевых срезов составляет 450-750 п.н. Дорожка M представляет собой маркер молекулярной массы (ДНК-лэддер NEB-50 п.н.), дорожка 1 представляет собой гель с HLA-Mix до разрезания, а дорожка 2 представляет собой гель с HLA-Mix после разрезания.
Фигура 8. Программа с захватом изображения на экране для конструирования консенсусной последовательности экзона 2 HLA-C-сайта образца No.2 в примере 8. Сначала последовательности-риды C-сайта образца сопоставляют путем выравнивания с эталонной последовательностью с помощью компьютерной программы BWA, затем конструируют консенсусные последовательности экзонов 2, 3, 4 С-сайта образца, после чего определяют последовательность гаплотипа каждого экзона C-сайта исходя из характера сцепления между SNP; и наконец, определяют тип образца по перекрыванию последовательностей экзонов этого гаплотипа. Как показано на этой фигуре, два гетерозиготных SNP находились в области 695-764 п.н. С-генной последовательности образца No.2, и исходя из считываемых последовательностей «рид 1» и «рид 2» можно определить, что характер сцепления SNP представляет собой A-C, G-A («…» на этой фигуре означает, что эти основания идентичны основаниям эталонной последовательности). Эти последовательности соответствуют заштрихованным частям последовательностей типа C*010201 и C*07020101, соответственно. Оценка характера сцепления в других областях давала аналогичные результаты.
Фигура 9. На этой фигуре проиллюстрированы результаты электрофореза ПЦР-продуктов для экзонов 2, 3, 4 HLA-C-сайта для 26 образцов примера 9. На этой фигуре все ПЦР-продукты имеют длину менее чем 500 п.н.; наблюдается одна электрофоретическая полоса, неспецифическая полоса отсутствует, а эффективности амплификации одной той же пары праймеров в различных образцах являются аналогичными.
Фигура 10. На этой фигуре проиллюстрированы результаты анализа данных секвенирования продуктов ПЦР-амплификации матрицы 1 с помощью компьютерной программы uType в примере 9. В столбце выходных данных слева приведены результаты *08:01:01 C*15:05:01, которые идентичны исходному известному результату для матрицы 1.
Фигура 11. На этой фигуре проиллюстрированы результаты электрофореза ПЦР-продуктов для экзонов 2+3 гена HLA-DQB1 в 94 образцах примера 10. На электрофореграмме видно, что ПЦР-продукты представляют собой серию отдельных полос размером 250 п.н. - 500 п.н., где дорожка M представляет собой эталон молекулярных масс стандартных ДНК (DL 2000, Takara Co.). Дорожки PI-1 - PI-94 представляют собой продукты ПЦР-амплификации экзонов 2+3 гена HLA-DQB1 в 94 образцах, а в негативном контроле (N) полоса амплификации отсутствует.
На фигуре 12 проиллюстрированы результаты электрофореза ДНК с разрезанием геля после фрагментирования HLA-Q-Mix, как описано в примере 10, где площадь гелевых срезов составляет 350-550 п.н. Дорожка M представляет собой маркер молекулярной массы стандартных ДНК (ДНК-лэддер NEB-50 п.н.), дорожка 1 представляет собой гель с HLA-Q-Mix до разрезания, а дорожка 2 представляет собой гель с HLA-Q-Mix после разрезания.
На фигуре 13 представлена программа с захватом изображения на экране для конструирования консенсусной последовательности образца No.7 в примере 10, иллюстрирующая главную процедуру анализа данных. Сначала считываемую последовательность DQB1-сайта образца выравнивают с эталонной последовательностью с помощью компьютерной программы BWA, затем конструируют консенсусные последовательности экзонов 2, 3 DQB1-образца, после чего определяют последовательность гаплотипов экзонов 2, 3 DQB1 исходя из характера сцепления между SNP. Как показано на этой фигуре, шесть гетерозиготных SNP находятся в области 2322-2412 последовательности гена DQB1 образца No.7; и исходя из считываемой последовательности «рид 1» можно определить, что характер сцепления SNP1-SNP5 составляет T-G-T-C-C; исходя из последовательности «рид 2» можно определить, что характер сцепления других SNP1-SNP5 составляет C-C-A-G-T; исходя из считываемой последовательности «рид 3» можно определить, что характер сцепления SNP3-SNP6 составляет A-G-T-G; исходя из считываемой последовательности «рид 4» можно определить, что характер сцепления других SNP3-SNP6 составляет T-C-C-A; а исходя из вышеуказанных характер сцепления для вышеуказанных SNP было определено, что считываемая последовательность «рид 1» сцеплена с последовательностью «рид 4», а считываемая последовательность «рид 2» сцеплена с последовательностью «рид 3», при этом полная комбинация SNP в этой области составляет T-G-T-C-C-A и C-C-A-G-T-G, и эти последовательности соответствуют заштрихованным частям последовательностей типа DQB1*0303 и DQB1*0602. Оценка характера сцепления в других областях давала аналогичные результаты.
На фигуре 14 представлена электрофореграмма продуктов примера 11, полученных после амплификации экзонов 2 и 3 HLA-DQB1-сайта и амплификации экзонов 2 и 3 с использованием двух пар ПЦР-праймеров, соответственно. На этой электрофореграмме показаны три серии ПЦР-продуктов от семи ДНК-матриц, где все ПЦР-продукты имеют длину менее чем 500 п.н., наблюдается одна электрофоретическая полоса, а неспецифическая полоса отсутствует. В негативной контроле (N) полоса амплификации отсутствует, а дорожка М представляет собой эталон молекулярных масс стандартных ДНК (DL 2000, Takara Co.).
На фигуре 15 проиллюстрированы результаты анализа данных секвенирования продуктов ПЦР-амплификации экзонов 2 и 3 HLA-DQB1 матрицы 7 с помощью компьютерной программы uType в примере 11. В столбце выходных данных слева приведены результаты DQB1*03:03 DQB1*06:02, которые идентичны исходному известному результату для матрицы 7.
На фигуре 16 представлены результаты электрофореза ПЦР-продуктов для соответствующих экзонов HLA-A/B/C/DQB1 в образце No.1 примера 12. На электрофореграмме видно, что ПЦР-продукты представляют собой серию отдельных полос размером 300 п.н. - 500 п.н., где дорожка M представляет собой маркер молекулярных масс (DL 2000, Takara Co.), дорожки 1-10 представляют собой продукты ПЦР-амплификации экзонов (A2, A3, A4, B2, B3, B4, C2, C3, C4, DQB1) HLA-A/B/C/DQB образца No.1, а в негативном контроле (N) полоса амплификации отсутствует. Результаты для других образцов являются аналогичными.
На фигуре 17 представлен результат выделения из агарозного геля после объединения в пул HLA-1-Mix, HLA-2-Mix, HLA-3-Mix, HLA-4-Mix, HLA-5-Mix, HLA-6-Mix, HLA-7-Mix, HLA-8-Mix, HLA-9-Mix и HLA-10-Mix в равном молярном соотношении, как описано в примере 12. Дорожка M представляет собой маркер молекулярных масс, дорожка 1 представляет собой результаты электрофоретического анализа пула, а дорожка 2 представляет собой электрофореграмму гелевых срезов, содержащих ДНК-фрагменты длиной 450-750 п.н.
На фигуре 18 представлена программа с захватом изображения на экране для конструирования консенсусной последовательности экзона 2 HLA-C-сайта образца No.1 в примере 12. Сначала считываемую последовательность C-сайта образца сопоставляют путем выравнивания с эталонной последовательностью с помощью компьютерной программы BWA, затем конструируют консенсусные последовательности экзонов 2, 3, 4 С-сайта образца, после чего определяют последовательности гаплотипа каждого экзона C-сайта исходя из характера сцепления между SNP; и наконец, определяют тип образца по перекрыванию последовательностей экзонов этого гаплотипа. Как показано на этой фигуре, два гетерозиготных SNP находились в области 695-764 С-генной последовательности образца No.1, и исходя из считываемых последовательностей «рид 1» и «рид 2» можно определить, что характер сцепления SNP представляет собой A-C, G-A («…» на этой фигуре означает, что эти основания идентичны основаниям эталонной последовательности). Эти последовательности соответствуют заштрихованным частям последовательностей типа C*010201 и C*07020101, соответственно. Оценка характера сцепления в других областях давала аналогичные результаты.
Примеры
Варианты настоящего изобретения подробно описаны в нижеследующих примерах. Однако для специалиста в данной области очевидно, что нижеследующие примеры приводятся лишь для иллюстрации настоящего изобретения и не должны рассматриваться как ограничение его объема.
В примерах 1-6 настоящего изобретения экзоны 2, 3, 4 HLA-A/B и экзон 2 HLA-DRB1 в 95 образцах были генотипированы с использованием комбинации праймерных индексов+стратегии неполного фрагментирования ДНК+парно-концевого метода секвенирования на секвенаторе Illumia GA 100 (ПЦР-продукты имели длину в пределах от 290 п.н. до 500 п.н.), что указывает на то, что такой способ согласно изобретению может быть применен для типирования генных фрагментов, длина которых превышает максимальную длину считываемых последовательностей в секвенаторе, который в достаточной степени удовлетворяет требованиям, предъявляемым к секвенатору второго поколения, таким как высокая производительность и низкая стоимость.
Принцип: для анализируемого образца, в два конца ПЦР-продуктов экзонов 2, 3, 4 HLA-A/B и экзона 2 HLA-DRB1 с помощью ПЦР-реакции вводили праймерные индексы в качестве специфических меток для получения информации о ПЦР-продуктах данного образца. Продукты ПЦР-амплификации трех сайтов (HLA-A/B/DRB1) в каждой группе образцов объединяли в пул с получением библиотеки ПЦР-продуктов, и после неполного ультразвукового фрагментирования библиотеки ПЦР-продуктов конструировали библиотеку для PCR-FREE-секвенирования. Полученную библиотеку для секвенирования подвергали электрофорезу в 2% агарозном геле с низкой температурой плавления, и все ДНК-полосы, длина которых составляла в пределах от 450 п.н. до 750 п.н., очищали и выделяли путем разрезания геля (поскольку адаптеры библиотеки добавляли к двум концам ДНК-фрагментов, то, во время конструирования библиотеки для PCR-FREE-секвенирования, длина ДНК-полосы, как было показано на электрофореграмме, примерно на 250 п.н. превышала фактическую длину ДНК-фрагментов, а поэтому выделенные здесь фрагменты, длина которых составляла в пределах от 450 п.н. до 700 п.н., фактически соответствовали ДНК-фрагментам, исходная длина которых составляла в пределах от 200 п.н. до 500 п.н.). Выделенную ДНК секвенировали на секвенаторе Illumina GA PE-100. Информация о последовательностях всех тестируемых образцов может быть получена благодаря последовательностям праймерных индексов, а последовательность полноразмерного ПЦР-продукта может быть получена путем сборки исходя из известных эталонных последовательностей и с учетом перекрывания и характера сцепления между последовательностями ДНК-фрагментов. Полноразмерная последовательность исходного ПЦР-продукта может быть получена путем сборки с использованием стандартной базы данных соответствующих экзонов HLA-A/B/DRB1, и последующего HLA-A/B/DRB1-генотипирования.
Пример 1
Экстракция образцов
ДНК экстрагировали из 95 проб крови с использованием известных результатов HLA-SBT-типирования (по Программе взятия костного мозга у доноров в Китае, далее называемой CMDP) на автоматическом устройстве для экстракции KingFisher (US Thermo Co.). При этом проводили следующие основные стадии: как описано в руководстве, определенное количество отдельно взятых агентов добавляли в шесть планшетов с глубокими лунками и в один планшет с неглубокими лунками с подсоединенным автоматическим оборудованием для экстракции KingFisher, и все планшеты, в которые были добавлены указанные агенты, помещали, если это необходимо, в соответствующие позиции. Была выбрана программа «Bioeasy_200ul Blood DNA_KF.msz», после чего запуск экстракции нуклеиновых кислот осуществляли путем нажатия кнопки «start». После завершения программы с планшета для элюирования было собрано приблизительно 100 мкл элюированных продуктов (то есть экстрагированной ДНК).
Пример 2
ПЦР-амплификация
Различные индексированные ПЦР-праймеры получали путем синтеза ПЦР-праймеров, имеющих различные праймерные индексы у 5'-конца, и такие различные индексированные ПЦР-праймеры могут быть нанесены на различные образцы, где ПЦР-праймерами являются ПЦР-праймеры для экзонов 2, 3, 4 HLA-A/B и экзона 2 HLA-DRB1. Затем праймерные индексы вводили в оба конца ПЦР-продуктов с помощью ПЦР-реакции для специфического мечения ПЦР-продуктов от различных образцов.
Для амплификации 95 образцов ДНК использовали 95 серий индексированных ПЦР-праймеров, соответственно, где каждая серия индексированных ПЦР-праймеров состояла из пары двунаправленных праймерных индексов (таблица 6) и ПЦР-праймеров для амплификации экзонов 2, 3, 4 HLA-A/B (таблица 1) и экзона 2 HLA-DRB1 (таблица 7), причем каждый прямой ПЦР-праймер имел индекс прямого праймера в паре праймерных индексов, присоединенных у 5'-конца, а обратный ПЦР-праймер имел индекс обратного праймера в паре праймерных индексов, присоединенных у 5′-конца. Во время синтеза праймеров эти праймерные индексы были непосредственно добавлены к 5′-концу ПЦР-праймеров.
95 ДНК, полученных после проведения стадии экстракции образцов, описанной в примере 1, обозначали как No. 1-95. ПЦР проводили в 96-луночных планшетах, всего в 7 планшетах, обозначенных HLA-P-A2, HLA-P-A3, HLA-P-A4, HLA-P-B2, HLA-P-B3, HLA-P-B4 и HLA-P-DRB1-2 (А2/А3/А4, В2/В3/В4, DRB1-2 представляют собой амплифицированные сайты), где негативный контроль в каждом планшете был добавлен без какой-либо матрицы, а праймеры, используемые в негативном контроле, были идентичны праймерам, используемым для матрицы 1. Во время проведения эксперимента регистрировали информацию о нумерации образцов, соответствующих каждой паре праймерных индексов.
53
54
55
56
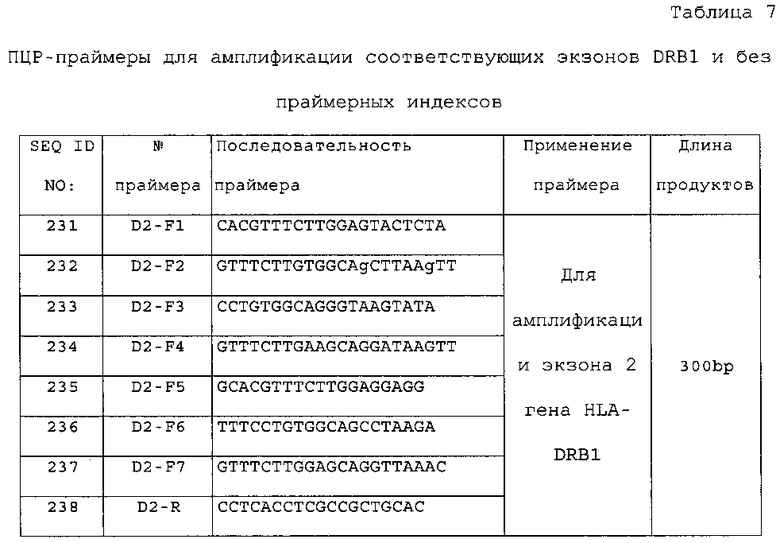
D2-F1, D2-F2, D2-F3, D2-F4, D2-F5, D2-F6, D2-F7 представляют собой прямые праймеры для амплификации экзона 2 HLA-DRB1, D2-R представляет собой обратный праймер для амплификации экзона 2 HLA-DRB1.
ПЦР для HLA-A/B/DRB1 проводили в следующих условиях:
96°С 2 мин
95°С 30 сек → 60°С 30 сек → 72°С 20 сек (32 цикла)
15°С ∞
ПЦР-реакцию для HLA-A/B проводили на нижеследующей системе, где все реагенты были закуплены у Promega (Beijing) Bio-Tech Co.
ПЦР-реакцию для HLA-DRB1 проводили по следующей системе:
Если PInf-A/B/D2-F1/2/3/4/5/6/7 представляет собой F-праймер HLA-A/B/DRB1, имеющий последовательность индекса прямого праймера No. n (таблица 6) у 5'-конца, то PInr-A/B/D2-R2/3/4 представляет собой R-праймер HLA-A/B/DRB1, имеющий последовательность индекса обратного праймера No. n у 5'-конца (в данном случае n≤95), а остальные праймеры могут быть получены аналогичным образом. Кроме того, каждый образец соответствует конкретной серии ПЦР-праймеров (PInf-A/B/D2-F1/2/3/4/5/6/7, PInr-A/B/D2-R2/3/4).
ПЦР-реакцию осуществляли на оборудовании для ПЦР PTC-200, поставляемом Bio-Rad Co. После проведения ПЦР 2 мкл ПЦР-продуктов подвергали электрофорезу в 1% агарозном геле. На фигуре 2 представлены результаты электрофореза ПЦР-продуктов соответствующих экзонов HLA-A/B/DRB1 образца No.1, а маркером молекулярных масс ДНК является DL 2000 (Takara Co.). На электрофореграмме наблюдалась серия одиночных полос длиной в пределах от 300 п.н. до 500 п.н., что указывало на успешную ПЦР-амплификацию экзонов (A2, A3, A4, B2, B3, B4, DRB1-2) HLA-A/B/DRB1 образца No.1. При этом в негативном контроле (N) полоса амплификации не наблюдалась. Результаты для других образцов были аналогичными.
Пример 3
Объединение в пул и очистка ПЦР-продуктов
20 мкл остальных ПЦР-продуктов, взятых из каждой лунки 96-луночного планшета с HLA-P-A2 (за исключением негативного контроля), смешивали со встряхиванием в 3-миллилитровой EP-пробирке до получения гомогенной смеси (обозначенной HLA-A2-Mix). Аналогичную процедуру проводили в других шести 96-луночных планшетах, обозначенных HLA-A3-Mix, HLA-A4-Mix, HLA-B2-Mix, HLA-B3-Mix, HLA-B4-Mix и HLA-D2-Mix. Из каждого планшета брали 200 мкл смеси HLA-A2-Mix, HLA-A3-Mix, HLA-A4-Mix, HLA-B2-Mix, HLA-B3-Mix, HLA-B4-Mix и HLA-D2-Mix и смешивали в 3-миллилитровой EP-пробирке, обозначенной HLA-Mix. 500 мкл ДНК-смеси от HLA-Mix подвергали очистке на колонке с использованием набора для очистки ДНК Qiagen (QIAGEN Co.) (Для подробного описания стадий очистки см. инструкции производителей). На оборудовании Nanodrop 8000 (Thermo Fisher Scientific Co.) было определено, что 200 мкл ДНК, полученной путем очистки, имеет концентрацию ДНК HLA-Mix, равную 48 нг/мкл.
Пример 4
Фрагментирование ПЦР-продуктов и конструирование библиотек для PCR-FREE-секвенирования на Illumina GA
1. Фрагментирование ДНК
Общее количество ДНК, составляющее 5 мкг и взятое из очищенной HLA-Mix, помещали в микропробирку Covaris с AFA-волокном, снабженную защелкивающейся крышкой, и подвергали фрагментированию на фрагментаторе Covaris S2DNA Shearer (Covaris Co.). Фрагментирование проводили в следующих условиях:
Качание частоты
2. Очистка после фрагментирования
Все фрагментированные продукты HLA-Mix выделяли и очищали с помощью набора для ПЦР-очистки QIAquick и растворяли в 37,5 мкл EB (элюирующего буфера QIAGEN), соответственно.
3. Реакция репарации концов
После фрагментации очищенную смесь HLA-Mix подвергали реакции репарации концов ДНК в следующей реакционной системе (все реагенты были закуплены у Enzymatics Co.):
Условия реакции: инкубирование при 20°C в течение 30 минут на термомиксере (Thermomixer, Eppendorf Co.).
Продукты реакции выделяли и очищали с помощью набора для ПЦР-очистки QIAquick и растворяли в 34 мкл EB (элюирующего буфера QIAGEN).
4. Добавление A у 3'-конца
A добавляли к 3'-концу ДНК, выделенной в последней стадии, с использованием следующей реакционной системы (все реагенты были закуплены у Enzymatics Co.):
Условия реакции: инкубирование при 37°C в течение 30 минут на термомиксере (Thermomixer, Eppendorf Co.).
Продукты реакции выделяли и очищали с помощью набора для ПЦР-очистки MiniElute (QIAGEN Co.) и растворяли в 13 мкл EB (элюирующего буфера QIAGEN).
5. Лигирование адаптера PCR-FREE-библиотеки на секвенаторе Illumina GA
Термин «адаптер PCR-FREE-библиотеки» означает сегмент, сконструированный из оснований, главная роль которых заключается в дополнительной фиксации молекулы ДНК на чипе для секвенирования и в обеспечении сайтов связывания для универсальных секвенирующих праймеров, где адаптер PCR-FREE-библиотеки может быть непосредственно лигирован с двумя концами ДНК-фрагментов в библиотеке для секвенирования, а поскольку при введении адаптера библиотеки ПЦР не проводят, то такой адаптер библиотеки называется здесь «адаптером PCR-FREE-библиотеки».
Продукты, имеющие добавленный A, лигировали с адаптерами PCR-FREE-библиотеки на Illumina GA с использованием следующей реакционной системы (все реагенты были закуплены у Illumina Co.):
Условия реакции: инкубирование при 20°C в течение 15 минут на термомиксере (Thermomixer, Eppendorf Co.).
Продукты реакции очищали на сферах Ampure (Beckman Coulter Genomics) и растворяли в 50 мкл деионизованной воды, а затем определяли концентрацию ДНК с помощью количественной ПЦР с активацией флуоресценции (QPCR) следующим образом:
кол. ПЦР (нМ)
6. Выделение путем разрезания геля
30 мкл HLA-Mix подвергали электрофорезу в 2% агарозном геле с низкой температурой плавления. Электрофорез проводили при 100 В, 100 минут. ДНК-маркер представлял собой 50 п.н. ДНК-маркера от NEB Co. Гель, содержащий ДНК-фрагменты размером от 450 до 750 п.н., разрезали (фигура 3). Продукты, выделенные из разрезанного геля, очищали с помощью набора для ПЦР-очистки QIAquick (QIAGEN Co.), и после такой очистки объем составлял 32 мкл, а концентрация ДНК, измеренная с помощью количественной ПЦР с активацией флуоресценции (QPCR), составляла 10,16 нМ.
Пример 5
Секвенирование на Illumina GA
В соответствии с результатами QPCR брали 10 пмоль ДНК и подвергали секвенированию по программе Illumina GA PE-100. Более подробное описание процедуры секвенирования см. в инструкции по секвенированию на Illumina GA (Illumina GA IIx).
Пример 6
Анализ результатов
Результаты секвенирования на Illumina GA представляют собой серию последовательностей ДНК, и после поиска последовательностей индексов прямого и обратного праймеров и последовательностей праймеров в результатах секвенирования создавали базы данных, содержащие результаты секвенирования ПЦР-продуктов различных экзонов HLA-A/B/DRB1 для каждого образца, соответствующего данному праймерному индексу. Результаты секвенирования каждого экзона сопоставляли путем выравнивания с эталонной последовательностью (эталонные последовательности можно найти на сайте http://www.ebi.ac.uk/imgt/hla/) соответствующего экзона с помощью программы BWA (Burrows-Wheeler Aligner), и таким образом конструировали консенсусные последовательности для каждой базы данных, а затем в этой базе данных отбирали последовательности ДНК и корректировали. Скорректированные последовательности ДНК получали путем их сборки в соответствующие последовательности экзонов HLA-A/B/DRB1 исходя из перекрывания последовательностей и характера сцепления (парно-концевого сцепления). Полученную последовательность ДНК сопоставляли путем выравнивания с последовательностью базы данных, соответствующей экзону HLA-A/B/DRB1 в профессиональной базе данных HLA IMGT. Если результат выравнивания последовательностей дает 100% соответствие, то это означает, что HLA-A/B/DRB1-генотип соответствующего образца был точно определен. См. программу с захватом изображения на экране для конструирования консенсусной последовательности экзона 2 HLA-A-сайта образца No.1, как показано на фигуре 4.
Для всех 95 образцов результаты типирования, полученные вышеописанным методом, полностью соответствовали известным результатам типирования, где результаты для образцов No.1-32 представлены ниже:



Примечание: в случае HLA типа HLA-DRB1, не исключено, что DRB1*1201 может представлять собой DRB1*1206/1210/1217, а DRB1*1454 может представлять собой DRB1*1401, поскольку указанные аллели полностью идентичны в последовательности экзона 2 гена HLA-DRB1.
Пример 7
Генотипирование HLA-A,B и DRB1 путем секвенирования на секвенаторе второго поколения (Illumina GA)
Экстракция образцов
ДНК экстрагировали из 950 проб крови с использованием известных результатов HLA-SBT-типирования (по Программе взятия костного мозга у доноров в Китае, далее называемой CMDP) на автоматическом оборудовании для экстракции KingFisher (US Thermo Co.). Этот метод описан в примере 1.
ПЦР-амплификация
950 ДНК, полученных после проведения каждой стадии экстракции образцов, были обозначены No.1-950, и были подразделены на 10 групп (95 ДНК для каждой группы), которые были обозначены как HLA-1, HLA-2, HLA-3, HLA-4, HLA-5, HLA-6, HLA-7, HLA-8, HLA-9, HLA-10. Для каждой группы образцов 95 образцов ДНК амплифицировали с использованием 95 наборов ПЦР-праймеров (таблица 1), несущих двунаправленные праймерные индексы (таблица 6), для амплификации экзонов 2, 3, 4 HLA-A/B и ПЦР-праймеров (таблица 7), несущих двунаправленные праймерные индексы (таблица 6), для амплификации экзона 2 HLA-DRB1. ПЦР-реакцию проводили в 96-луночных планшетах с использованием всего 70 планшетов, обозначенных HLA-X-P-A2, HLA-X-P-A3, HLA-X-P-A4, HLA-X-P-B2, HLA-X-P-B3, HLA-X-P-B4 и HLA-X-P-DRB1-2 («X» указывает на номер группы 1/2/3/4/5/6/7/8/9/10, а «A2/3/4», «B2/3/4», «DRB1-2» представляют собой сайты амплификации), где в каждом планшете имеется негативный контроль без какой-либо матрицы, а праймеры, используемые для негативного контроля, представляют собой праймеры, помеченные PI-1 (таблица 6). Во время эксперимента регистрировали номер каждого образца в группе и информацию о праймерных индексах. Этот способ описан в примере 2.
Объединение в пул и очистка ПЦР-продуктов
Для образцов группы X («X» представляет собой 1/2/3/4/5/6/7/8/9/10), 20 мкл остальных ПЦР-продуктов, взятых из каждой лунки 96-луночного планшета с HLA-Х-P-A2 (за исключением негативного контроля), смешивали со встряхиванием в 3-миллилитровой EP-пробирке до получения гомогенной смеси (обозначенной HLA-Х-A2-Mix). Аналогичную процедуру проводили в других шести 96-луночных планшетах с образцами группы Х, обозначенных HLA-X-A3-Mix, HLA-X-A4-Mix, HLA-X-B2-Mix, HLA-X-B3-Mix, HLA-X-B4-Mix и HLA-X-D2-Mix. Из каждого планшета брали 200 мкл смеси HLA-X-A2-Mix, HLA-X-A3-Mix, HLA-X-A4-Mix, HLA-X-B2-Mix, HLA-X-B3-Mix, HLA-X-B4-Mix и HLA-X-D2-Mix, и смешивали в 3-миллилитровой EP-пробирке, обозначенной HLA-Х-Mix. 500 мкл ДНК-смеси от HLA-Х-Mix подвергали очистке на колонке с использованием набора для очистки ДНК Qiagen (QIAGEN Co.) (Для подробного описания стадий очистки см. инструкции производителей) с получением 200 мкл ДНК, концентрацию которой определяли на оборудовании Nanodrop 8000 (Thermo Fisher Scientific Co.). Ту же самую процедуру проводили и для других групп. Конечные определенные концентрации ДНК указаны ниже.
Mix
Mix
Этот метод описан в примере 3.
Конструирование библиотек для секвенирования осуществляли на Illumina GA методом, описанным в примере 4. Ниже показана соответствующая взаимосвязь между группами образцов и адаптерами библиотеки.
Продукты реакции очищали на сферах Ampure (Beckman Coulter Genomics) и растворяли в 50 мкл деионизованной воды, а затем определяли молярные концентрации ДНК с помощью количественной ПЦР с активацией флуоресценции (QPCR) следующим образом:
Выделение путем разрезания геля
HLA-1-Mix, HLA-2-Mix, HLA-3-Mix, HLA-4-Mix, HLA-5-Mix, HLA-6-Mix, HLA-7-Mix, HLA-8-Mix, HLA-9-Mix и HLA-10-Mix смешивали в равных молярных концентрациях (конечная концентрация составляла 72,13 нМ/мкл) и обозначали HLA-Mix-10. 30 мкл HLA-Mix-10 подвергали электрофорезу в 2% агарозном геле с низкой температурой плавления. Электрофорез проводили при 100 В, 100 минут. ДНК-маркер представлял собой 50 п.н. ДНК-маркера от NEB Co. Гель, содержащий ДНК-фрагменты размером от 450 до 750 п.н., разрезали. Продукты, выделенные из разрезанного геля, очищали с помощью набора для ПЦР-очистки QIAquick (QIAGEN Co.), и после такой очистки объем составлял 32 мкл, а концентрация ДНК, измеренная с помощью количественной ПЦР с активацией флуоресценции (QPCR) составляла 9,96 нМ.
Секвенирование и анализ результатов осуществляли, как описано в примерах 5 и 6. Для всех 950 образцов результаты типирования, полученные вышеописанным методом, полностью соответствовали известным результатам типирования.
Пример 8
HLA-C-генотипирование с применением метода секвенирования на секвенаторе второго поколения (Illumina GA)
1. Экстракция ДНК-образцов
Стадии проводили, как описано в примере 1.
2. ПЦР-амплификация
Стадии проводили, как описано в примере 2, за исключением того, что используемыми ПЦР-праймерами являются ПЦР-праймеры для экзонов 2, 3 и 4 HLA-C, как показано в таблице 3.
Для амплификации 95 образцов ДНК использовали 95 серий индексированных ПЦР-праймеров, соответственно, где каждая серия индексированных ПЦР-праймеров состояла из ПЦР-праймеров для амплификации экзонов 2, 3, 4 HLA-С (таблица 3) и пары двунаправленных праймерных индексов (как описано ниже), где каждый прямой ПЦР-праймер имел индекс прямого праймера в паре праймерных индексов, присоединенных у 5'-конца, а обратный ПЦР-праймер имел индекс обратного праймера в паре праймерных индексов, присоединенных у 5'-конца. Во время синтеза праймеров, эти праймерные индексы были непосредственно добавлены к 5'-концу ПЦР-праймеров.
95 ДНК, полученных после проведения стадии экстракции образцов, обозначали No. 1-95. ПЦР проводили в 96-луночных планшетах, всего в 3 планшетах, обозначенных HLA-P-C2, HLA-P-C3, HLA-P-A4 (С2/3/4 представляют собой сайты амплификации), где негативный контроль в каждом планшете был добавлен без какой-либо матрицы, а праймеры, используемые в негативном контроле, были идентичны праймерам PI-96. Во время проведения эксперимента регистрировали информацию о нумерации образцов, соответствующих каждой паре праймерных индексов.
Используемыми праймерными индексами являются праймерные индексы PI-1 - PI-95, перечисленные в таблице 6, и указанный ниже праймерный индекс PI-96, используемый в качестве негативного контроля (таблица 8)

ДНК, экстрагированные на автоматическом оборудовании для экстракции KingFisher в стадии 1, использовали в качестве матриц и ПЦР-амплификацию осуществляли в отдельных пробирках с использованием праймеров для экзонов HLA-C, где праймеры были индексированы у 5′-конца. ПЦР проводили следующим образом:
С2: 96°С, 2 мин
95°С 30 сек → 62°С 30 сек → 72°С 20 сек (35 циклов)
15°С ∞
C3: 96°C 2 мин
95°C 30 сек → 56°C 30 сек → 72°C 20 сек (35 циклов)
15°C ∞
C4: 96 °C 2 мин
95°C 30 сек → 60°C 30сек → 72°C 20 сек (35 циклов)
15°C ∞
ПЦР-реакцию для HLA-С проводили на нижеследующей системе:
Если PInf-C-F2/3/4 представляет собой F-праймер HLA-С, имеющий последовательность индекса прямого праймера No. n (таблица 2) у 5'-конца, то PInr-C-R2/3/4 представляет собой R-праймер HLA-С, имеющий последовательность индекса обратного праймера No. n у 5'-конца (в данном случае n≤96), а остальные праймеры могут быть получены аналогичным образом. Кроме того, каждый образец соответствует конкретной серии ПЦР-праймеров.
ПЦР-реакцию осуществляли на оборудовании для ПЦР PTC-200, поставляемом Bio-Rad Co. После проведения ПЦР 2 мкл ПЦР-продуктов подвергали электрофорезу в 1,5% агарозном геле. На фигуре 6 представлены результаты электрофореза ПЦР-продуктов соответствующих экзонов HLA-С первых 20 образцов, а маркером молекулярных масс ДНК является DL 2000 (Takara Co.). На электрофореграмме наблюдалась серия одиночных полос длиной в пределах от 400 п.н. до 500 п.н., что указывало на успешную ПЦР-амплификацию экзонов (С2, С3, С4) HLA-С-образцов. Результаты для других образцов были аналогичными.
Объединение в пул и очистка ПЦР-продуктов
20 мкл остальных ПЦР-продуктов, взятых из каждой лунки 96-луночного планшета с HLA-P-С2 (за исключением негативного контроля), объединяли в гомогенный пул со встряхиванием в 3-миллилитровой EP-пробирке (обозначенный HLA-С2-Mix). Аналогичную процедуру проводили в других двух 96-луночных планшетах, обозначенных HLA-С3-Mix и HLA-С4-Mix. Из каждого планшета брали 200 мкл каждого из HLA-С2-Mix, HLA-С3-Mix и HLA-С4-Mix, и смешивали в 1,5-миллилитровой EP-пробирке, обозначенной HLA-Mix. 500 мкл ДНК-смеси от HLA-Mix подвергали очистке на колонке с использованием набора для очистки ДНК Qiagen (QIAGEN Co.) (Для подробного описания стадий очистки см. инструкции производителей). На оборудовании Nanodrop 8000 (Thermo Fisher Scientific Co.) было определено, что 200 мкл ДНК, полученной путем очистки, имеет концентрацию ДНК HLA-Mix, равную 50 нг/мкл.
4. Конструирование библиотек для PCR-FREE-секвенирования на Illumina GA
4.1. Фрагментирование ПЦР-продуктов
Общее количество ДНК, составляющее 5 мкг и взятое из очищенной HLA-Mix, помещали в микропробирку Covaris с AFA-волокном, снабженную защелкивающейся крышкой, и подвергали фрагментированию на фрагментаторе Covaris S2 (Covaris Co.). Фрагментирование проводили в следующих условиях:
Качание частоты
4.2. Очистка фрагментированных ПЦР-продуктов
Все фрагментированные продукты HLA-Mix выделяли и очищали с помощью набора для ПЦР-очистки QIAquick и растворяли в 37,5 мкл EB (элюирующего буфера QIAGEN), соответственно.
4.3. Реакция репарации концов
Очищенные продукты подвергали реакции репарации концов ДНК в в следующей реакционной системе (все реагенты были закуплены у Enzymatics Co.):
Условия реакции: инкубирование при 20ºC в течение 30 минут на термомиксере (Thermomixer, Eppendorf Co.).
Продукты реакции выделяли и очищали с помощью набора для ПЦР-очистки QIAquick и растворяли в 32 мкл EB (элюирующего буфера QIAGEN).
4.4. Добавление A у 3'-конца
A добавляли к 3'-концу ДНК, выделенной в последней стадии, с использованием следующей реакционной системы (все реагенты были закуплены у Enzymatics Co.):
Условия реакции: инкубирование при 37°C в течение 30 минут в термомиксере (Thermomixer, Eppendorf Co.).
Продукты реакции выделяли и очищали с помощью набора для ПЦР-очистки MiniElute (QIAGEN Co.) и растворяли в 38 мкл EB (элюирующего буфера QIAGEN).
4.5. Лигирование адаптера PCR-FREE-библиотеки на секвенаторе Illumina GA
Продукты, имеющие добавленный A, лигировали с адаптерами PCR-FREE-библиотеки на Illumina GA с использованием следующей реакционной системы (все реагенты были закуплены у Illumina Co.):
Условия реакции: инкубирование при 16°C в течение ночи в термомиксере (Thermomixer, Eppendorf Co.).
Продукты реакции очищали на сферах Ampure (Beckman Coulter Genomics) и растворяли в 50 мкл деионизованной воды, а затем определяли концентрацию ДНК с помощью количественной ПЦР с активацией флуоресценции (QPCR) следующим образом:
4.6. Выделение путем разрезания геля
30 мкл HLA-Mix подвергали электрофорезу в 2% агарозном геле с низкой температурой плавления. Электрофорез проводили при 100 В, 100 минут. ДНК-маркер представлял собой 50 п.н. ДНК-лэддера от NEB Co. Гель, содержащий ДНК-фрагменты размером от 450 до 750 п.н., разрезали (фигура 7). Продукты, выделенные из разрезанного геля, очищали с помощью набора для ПЦР-очистки QIAquick (QIAGEN Co.), и после такой очистки объем составлял 32 мкл, а концентрация ДНК, измеренная с помощью количественной ПЦР с активацией флуоресценции (QPCR), составляла 17,16 нМ.
5. Секвенирование на Illumina GA
В соответствии с результатами QPCR-детектирования брали 10 пмоль ДНК и подвергали секвенированию по программе Illumina GA PE-100. Более подробное описание процедуры секвенирования см. в инструкции по секвенированию на Illumina GA (Illumina GA IIx).
6. Анализ результатов
Результаты секвенирования, полученные на Illumina GA, представляют собой серию последовательностей ДНК, и после поиска последовательностей индексов прямого и обратного праймеров и последовательностей праймеров в этих результатах секвенирования создавали базы данных, содержащие результаты секвенирования ПЦР-продуктов различных экзонов HLA-С для каждого образца, соответствующего данному праймерному индексу. Результаты секвенирования каждого экзона сопоставляли путем выравнивания с эталонной последовательностью (эталонные последовательности можно найти на сайте http://www.ebi.ac.uk/imgt/hla/) соответствующего экзона с помощью программы BWA (Burrows-Wheeler Aligner), и таким образом конструировали консенсусные последовательности для каждой базы данных, а затем в этой базе данных отбирали последовательности «риды» и корректировали исходя из качественной оценки секвенирования оснований и различия между последовательностями-«ридами» и консенсусными последовательностями. Скорректированные последовательности ДНК получали путем сборки в соответствующие последовательности экзонов HLA-С исходя из перекрывания последовательностей и характера сцепления (парно-концевого сцепления). Процедура конструирования консенсусных последовательностей экзона 2 HLA-С-сайта в образце № 2, проводимая с помощью программы с захватом изображения на экране, проиллюстрирована на фигуре 8.
Полученную ДНК-последовательность сопоставляли с последовательностями базы данных для соответствующего экзона HLA-С, имеющегося в профессиональной базе данных HLA IMGT. Если результат выравнивания последовательностей давал 100% соответствие, то это означает, что HLA-С-генотип соответствующего образца был точно определен. Для всех 95 образцов результаты типирования, полученные вышеописанным методом, полностью соответствовали известным результатам типирования, где результаты типирования для образцов No.1-32 представлены ниже (как видно из таблицы 9, все полученные результаты были идентичны исходным известным результатам):
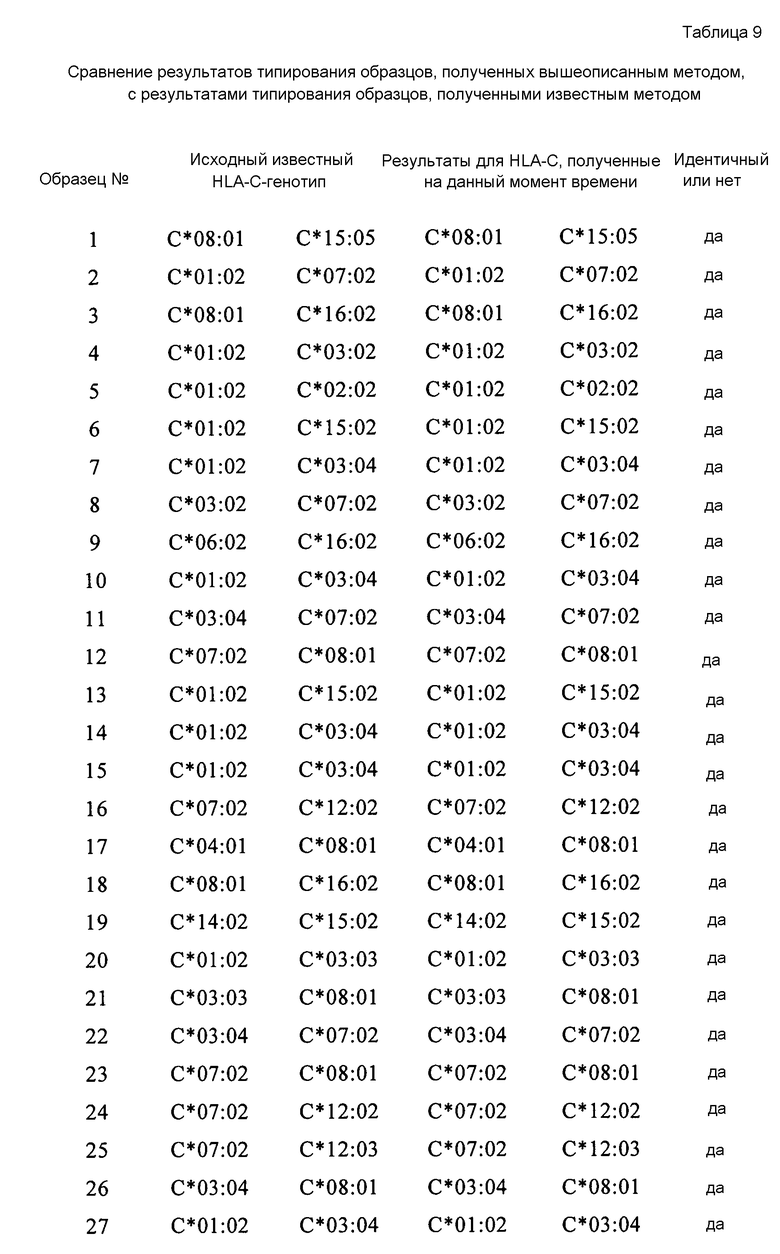

Примечание: в случае HLA типа HLA-С, не исключено, что C*0303 может представлять собой C*0320N, C*0401 может представлять собой C*0409N/0430, C*0702 может представлять собой C*0750, C*0801 может представлять собой C*0822, а C*1505 может представлять собой C*1529, поскольку указанные аллели полностью идентичны в последовательностях экзонов 2, 3, 4 HLA-C.
Пример 9
HLA-С-генотипирование методом секвенирования по Сэнгеру
1. Экстракция образцов ДНК
Как описано в примере 1, на автоматическом оборудовании для экстракции KingFisher экстрагировали 26 из 95 образцов ДНК с известными HLA-генотипами.
2. ПЦР-амплификация
Вышеуказанные ДНК, экстрагированные на автоматическом оборудовании для экстракции KingFisher, использовали в качестве матриц и ПЦР-амплификацию осуществляли в отдельных пробирках с использованием трех пар ПЦР-праймеров C-F2/C-R2, C-F3/C-R3, C-F4/C-R4 (таблица 3), соответственно. Для каждой пары праймеров проводили ПЦР в следующих условиях:
C2: 96°C, 2 мин
95°C 30 сек → 62°C 30 сек → 72°C 20 сек (35 циклов)
15°C ∞
C3: 96°C 2 мин
95°C 30 сек → 56°C 30 сек → 72°C 20 сек (35 циклов)
15°C ∞
C4: 96°C 2 мин
95°C 30 сек → 60°C 30 сек → 72°C 20 сек (35 циклов)
15°C ∞
ПЦР-реакцию для HLA-С проводили на нижеследующей системе:
Перед очисткой ПЦР-продукты подвергали электрофорезу в агарозном геле (фигура 9).
3. Очистка ПЦР-продуктов
ПЦР-продукты очищали на планшетах для очистки Millipore. Были проведены следующие основные стадии. Используемые лунки 96-луночного планшета для очистки ПЦР-продуктов помечали маркером и в каждую используемую лунку добавляли 50 мкл сверхчистой воды. Остальные лунки герметично заклеивали пленкой-герметиком. Планшет оставляли на 15 минут или подсоединяли на 5 минут к дренажной и фильтрующей системе (~10 Па). При отсоединении планшета для очистки от дренажной и фильтрующей системы, жидкость, находящуюся в съемном порте на дне планшета для очистки, удаляли с помощью влагопоглощающей бумаги.
Предназначенные для очистки ПЦР-продукты центрифугировали в течение 1 минуты при 4000 об/мин, покрытие или слой силикагеля для очищаемых ПЦР-продуктов удаляли и в каждую систему для ПЦР-реакции добавляли 100 мкл сверхчистой воды. Затем планшет для очистки, в который были добавлены очищаемые ПЦР-продукты, подсоединяли к дренажной и фильтрующей системе и уровень вакуума доводили до ~10 Па, на что указывал барометр. Дренаж и фильтрование продолжали до тех пор, пока с микропористой регенерируемой целлюлозной пленки, находящейся на дне планшета для очистки, не исчезала жидкость и не пропадал блеск, наблюдаемый в результате отражения света от интактной поверхности жидкости.
В лунках, содержащих очищаемые ПЦР-продукты, в микропористую регенерируемую целлюлозную пленку добавляли 50 мкл сверхчистой воды или TE, а затем планшет для очистки подвергали встряхиванию при комнатной температуре в шейкере-самописце в течение 5 минут и всю жидкость, содержащуюся в соответствующих лунках, переносили в соответствующие лунки нового 96-луночного ПЦР-планшета.
4. Осуществление реакции секвенирования и очистка продуктов реакции секвенирования
ПЦР-продукты, очищенные, как описано выше, использовали в качестве матриц для реакции секвенирования.
Условия проведения реакции секвенирования:
96°C 2 мин
96°C 10 сек → 55°C 5 сек → 60°C 2 мин (25 циклов)
15°C ∞
Система для реакции секвенирования
Продукты реакции секвенирования очищали путем проведения следующих стадий: планшет для реакции секвенирования уравновешивали и центрифугировали при 3000×g в течение 1 минуты. В 96-луночном планшете к каждой реакционной 5-мкл системе добавляли 2 мкл 0,125 моль/л EDTA-Na2-раствора, 33 мкл 85% этанола и планшет покрывали слоем силикагеля, а затем достаточно интенсивно встряхивали в течение 3 минут. Затем планшет центрифугировали при 4°C, 3000×g в течение 30 минут. После центрифугирования планшет для секвенирования вынимали, слой силикагеля удаляли и планшет для секвенирования помещали верхом вниз на влагопоглощающую бумагу, после чего подвергали инверсионному центрифугированию до тех пор, пока центрифужная сила не достигала 185×g. Затем в каждую лунку 96-луночного планшета добавляли 50 мкл 70% этанола. Планшет покрывали слоем силикагеля, встряхивали в течение 1,5 минут и центрифугировали при 4°C, 3000×g в течение 15 минут. Затем планшет для реакции секвенирования помещали на 30 минут в темное и проветриваемое помещение и сушили воздухом до тех пор, пока не исчезал запах этанола. В каждую лунку 96-луночного планшета добавляли 10 мкл HI-DI-формамида (альтернативно, в каждую лунку 384-луночного планшета добавляли 8 мкл HI-DI-формамида), а затем этот планшет закрывали пленкой-герметиком и после встряхивания в течение 5 секунд центрифугировали при 1000 об/мин.
5. Секвенирование и анализ результатов
Очищенные продукты реакции секвенирования подвергали капиллярному электрофорезу в секвенирующем геле ABI 3730XL. Пики секвенирования анализировали с помощью компьютерной программы uType (Invitrogen) и получали результаты HLA-типирования (фигура 10). Все результаты, полученные вышеописанным методом, были идентичны первоначальным известным результатам, как показано в таблице 10.
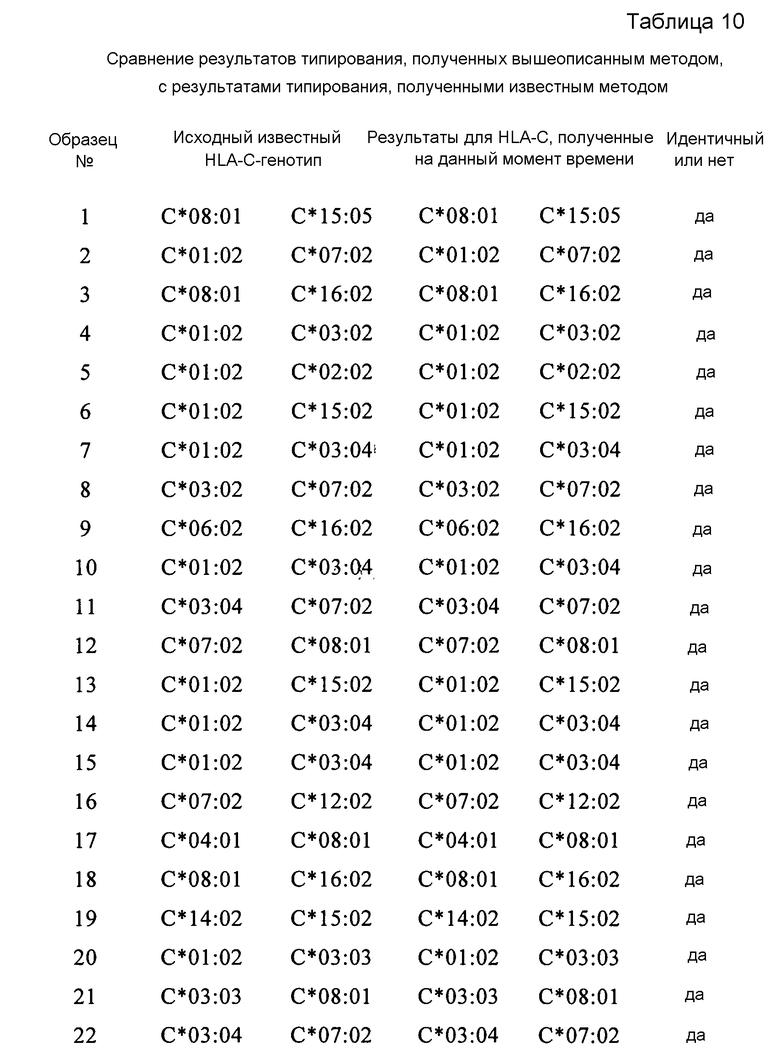

Пример 10: HLA-DQB1-генотипирование методом секвенирования второго поколения (Illumina Solexa)
94 пробы крови с известными результатами HLA-SBT-типирования подвергали HLA-DQB1-генотипированию методами, описанными в примере 8, за исключением нижеследующих изменений, представленных ниже.
Для амплификации 94 образцов ДНК использовали 94 серии индексированных ПЦР-праймеров, соответственно, где каждая серия индексированных ПЦР-праймеров состояла из ПЦР-праймеров для амплификации экзонов 2 или 3 HLA-DQB1 (таблица 5) и пары двунаправленных праймерных индексов (как описано выше), где каждый прямой ПЦР-праймер имел индекс прямого праймера в паре праймерных индексов, присоединенных у 5'-конца, а обратный ПЦР-праймер имел индекс обратного праймера в паре праймерных индексов, присоединенных у 5'-конца. Во время синтеза праймеров, эти праймерные индексы были непосредственно добавлены к 5'-концу ПЦР-праймеров, где указанные праймеры были синтезированы фирмой Shanghai Invitrogen Co.
94 ДНК, полученных после проведения стадии экстракции образцов, обозначали No.1-94. ПЦР проводили в 96-луночных планшетах, где в одной и той же лунке были амплифицированы экзоны 2, 3 DQB1 в каждом образце. Два негативных контроля в каждом планшете были добавлены без какой-либо матрицы, а праймерными индексами, используемыми в качестве негативного контроля, являются PI-95 и PI-96. Во время проведения эксперимента регистрировали данные о нумерации образцов, соответствующих каждой паре праймерных индексов.
В качестве праймерных индексов использовали праймерные индексы PI-1 - PI-94, перечисленные в таблице 6, а указанные ниже праймерные индексы PI-95 и PI-96 (таблица 11) были использованы для негативного контроля.

ПЦР-процедура для HLA-DQB1:
96°С 2 мин
95°С 30 сек → 60°С 30 сек → 72°С 20 сек (32 цикла)
15°С ∞
ПЦР-реакцию для HLA-DQB1 проводили на нижеследующей системе:
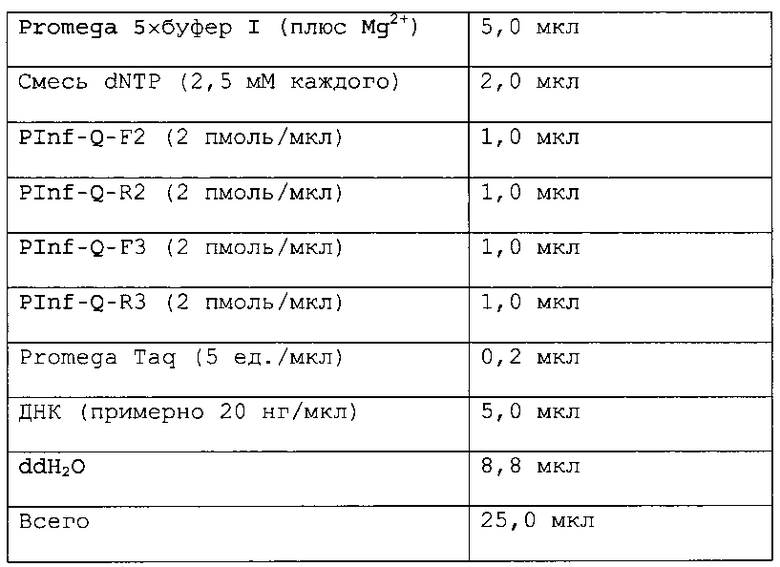
Если PInf-Q-F2/3 представляет собой F-праймер HLA-DQB1, имеющий последовательность индекса прямого праймера No. n
(таблица 1) у 5'-конца, то PInf-Q-R2/3 представляет собой R-праймер HLA-DQB1, имеющий последовательность индекса обратного праймера No. n у 5'-конца (в данном случае n≤96), а остальные праймеры могут быть получены аналогичным образом. Кроме того, каждый образец соответствует конкретной серии ПЦР-праймеров.
ПЦР-реакцию осуществляли на оборудовании для ПЦР PTC-200, поставляемом Bio-Rad Co. После проведения ПЦР 2 мкл ПЦР-продуктов подвергали электрофорезу в 1,5% агарозном геле. На фигуре 11 представлены результаты электрофореза ПЦР-продуктов экзонов 2+3 HLA-DQB1 для 94 образцов, а маркером молекулярных масс ДНК является DL 2000 (Takara Co.).
Объединение в пул и очистка ПЦР-продуктов
20 мкл остальных ПЦР-продуктов, взятых из каждой лунки 96-луночного планшета с HLA-P-DQB1 (за исключением негативного контроля), смешивали до гомогенности в пул в 3-миллилитровой EP-пробирке (обозначенной HLA-Q-Mix). 500 мкл ДНК-смеси от HLA-Q-Mix подвергали очистке на колонке с использованием набора для очистки ДНК Qiagen (QIAGEN Co.) (Для подробного описания стадий очистки см. инструкции производителей). На оборудовании Nanodrop 8000 (Thermo Fisher Scientific Co.) было определено, что 200 мкл ДНК, полученной путем очистки, имеет концентрацию ДНК HLA-Q-Mix, равную 48 нг/мкл.
Фрагментирование проводили в следующих условиях:
Качание частоты
Продукты реакции подвергали реакции репарации концов, а затем выделяли и очищали с помощью набора для ПЦР-очистки QIAquick, и растворяли в 34 мкл EB (элюирующего буфера QIAGEN).
Затем к продуктам реакции присоединяли А у 3'-конца, после чего эти продукты выделяли и очищали с помощью набора для ПЦР-очистки MiniElute (QIAGEN Co.) и растворяли в 13 мкл раствора EB (элюирующего буфера QIAGEN).
После лигирования адаптеров библиотеки, продукты реакции очищали на сферах Ampure (Beckman Coulter Genomics) и растворяли в 50 мкл деионизованной воды, а затем определяли концентрацию ДНК с помощью количественной ПЦР с активацией флуоресценции (QPCR) следующим образом:
Гель, содержащий ДНК-фрагменты размером от 350 до 550 п.н., разрезали (фигура 12). После очистки и выделения продуктов из геля, концентрация ДНК, измеренная с помощью количественной ПЦР с активацией флуоресценции (QPCR), составляла 18,83 нМ.
Анализ результатов
Результаты секвенирования, полученные на Illumina GA, представляют собой серию последовательностей ДНК, и после поиска последовательностей индексов прямого и обратного праймеров и последовательностей праймеров в результатах секвенирования создавали базы данных, содержащие результаты секвенирования ПЦР-продуктов различных экзонов HLA-DQB1 для каждого образца, соответствующего данному праймерному индексу. Результаты секвенирования каждого экзона сопоставляли путем выравнивания с эталонной последовательностью (эталонные последовательности можно найти на сайте http://www.ebi.ac.uk/imgt/hla/) соответствующего экзона с помощью программы BWA (Burrows-Wheeler Aligner), и таким образом конструировали консенсусные последовательности для каждой базы данных, а затем в этой базе данных отбирали последовательности «ридов» и корректировали исходя из качественной оценки секвенирования оснований и различий между последовательностями «ридов» и консенсусными последовательностями. Скорректированные последовательности ДНК получали путем сборки в соответствующие последовательности экзонов 2,3 HLA-DQB1 исходя из перекрывания и характера сцепления (парно-концевого сцепления) последовательностей. Процедура конструирования консенсусной последовательности экзона 2 HLA-DQB1-сайта в образце no.7, проводимая с помощью программы с захватом изображения на экране, проиллюстрирована на фигуре 13.
Полученную ДНК-последовательность для экзонов 2,3 HLA-DQB1 сопоставляли с последовательностями базы данных для соответствующего экзона HLA-DQB1, имеющегося в профессиональной базе данных HLA IMGT. Если результат выравнивания последовательностей дает 100% соответствие, то это означает, что HLA-DQB1-генотип соответствующего образца был точно определен.
Для всех 94 образцов результаты типирования, полученные вышеописанным методом, полностью соответствовали известным результатам типирования, где результаты для образцов No.1-32 представлены в таблице 12.
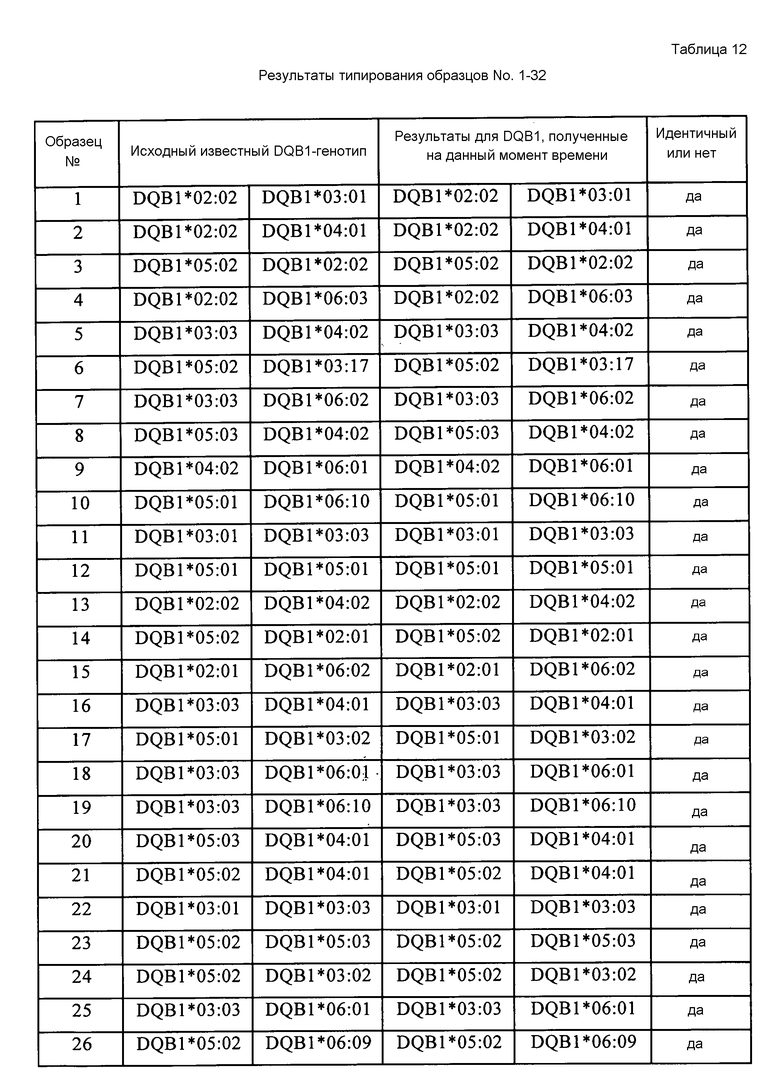
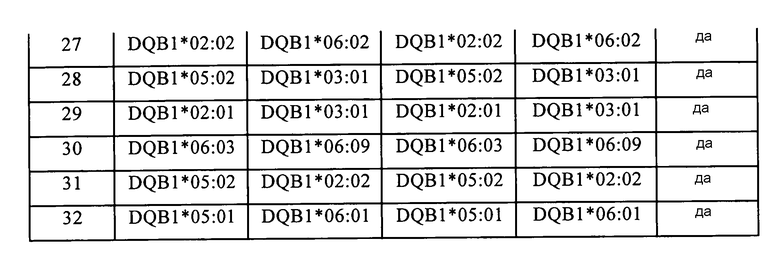
Пример 11: HLA-DQB1-генотипирование методом секвенирования по Сэнгеру
1. Экстракция образцов ДНК
Как описано в примере 1, на автоматическом оборудовании для экстракции KingFisher экстрагировали 20 из 94 образцов ДНК с известными HLA-генотипами.
2. ПЦР-амплификация
Вышеуказанные ДНК, экстрагированные на автоматическом оборудовании для экстракции KingFisher, использовали в качестве матриц и ПЦР-амплификацию осуществляли в отдельных пробирках с использованием двух пар ПЦР-праймеров (Q-F2 и Q-R2, Q-F3 и Q-R3), перечисленных в таблице 5, соответственно. Для каждой пары праймеров проводили ПЦР в следующих условиях:
96°C 2 мин
95°C 30 сек → 56°C 30 сек → 72°С 20 сек (35 циклов)
15°C ∞
ПЦР-реакцию для HLA-Q проводили на нижеследующей системе:
Перед очисткой ПЦР-продукты подвергали электрофорезу в агарозном геле.
3. Очистка ПЦР-продуктов
Очистку проводили с применением метода и стадий, описанных в примере 9.
4. Осуществление реакции секвенирования и очистки продуктов реакции секвенирования
Эту реакцию и очистку проводили с применением метода и стадий, описанных в примере 9.
5. Секвенирование и анализ результатов
Очищенные продукты реакции секвенирования подвергали капиллярному электрофорезу в секвенирующем геле ABI 37 30XL. Пики секвенирования анализировали с помощью компьютерной программы uType (Invitrogen) и получали результаты HLA-типирования (фигура 15). Все результаты, полученные вышеописанным методом, были идентичны первоначальным известным результатам, как показано в таблице 13.
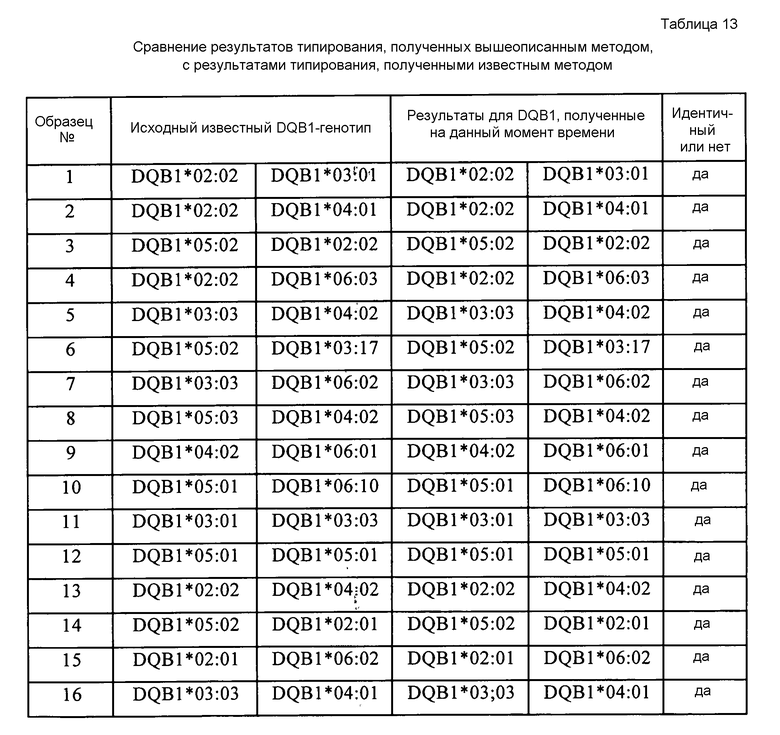
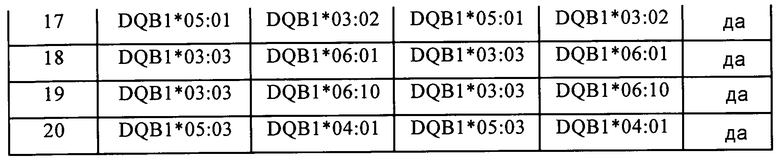
Пример 12: Генотипирование экзонов 2, 3, 4 HLA-A/B/C и экзонов 2, 3 HLA-DQB1 в 950 образцах
В примерах настоящего изобретения экзоны 2, 3, 4 HLA-A/B/С и экзоны 2,3 HLA-DQB1 в 950 образцах были генотипированы с помощью комбинации праймерных индексов, стратегии неполного фрагментирования ДНК, индексов библиотеки, получения PCR-FREE-библиотек и парно-концевого метода секвенирования на секвенаторе Illumia GA 100 (ПЦР-продукты имели длину в пределах от 300 п.н. до 500 п.н.), что указывает на то, что такой способ согласно изобретению может быть применен для генотипирования генных фрагментов, длина которых превышает максимальную длину «ридов» в секвенаторе, и что настоящее изобретение позволяет осуществлять недорогостоящее HLA-генотипирование с высокой производительностью, высокой точностью и высоким разрешением.
Принцип: анализируемые образцы разделяли на 10 групп, и в образцах каждой группы в два конца ПЦР-продуктов экзонов 2, 3, 4 HLA-A/B/С и экзонов 2,3 HLA-DQB1 с помощью ПЦР-реакции вводили праймерные индексы в качестве специфических меток для получения информации о ПЦР-продуктах данного образца. Продукты ПЦР-амплификации четырех сайтов (HLA-A/B/С/DQB1) в каждой группе образцов смешивали с получением библиотеки ПЦР-продуктов, и после неполного ультразвукового фрагментирования библиотек ПЦР-продуктов конструировали индексированные PCR-FREE-библиотеки для секвенирования (если для библиотеки ПЦР-продуктов в образце каждой группы использовали различные адаптеры, то было сконструировано 10 индексированных библиотек для секвенирования). 10 индексированных библиотек для секвенирования объединяли в пул в равной молярной концентрации и получали смешанную индексированную библиотеку для секвенирования. Эту смешанную индексированную библиотеку для секвенирования подвергали электрофорезу в 2% агарозном геле с низкой температурой плавления, и все ДНК-полосы, длина которых составляла в пределах от 450 п.н. до 750 п.н., очищали и выделяли путем разрезания геля. Выделенную ДНК секвенировали методом Illumina GA PE-100. Информация о последовательностях всех тестируемых образцов может быть получена исходя из последовательностей праймерных индексов и последовательностей индексов библиотеки, а последовательность полноразмерного ПЦР-продукта может быть получена путем сборки исходя из известных эталонных последовательностей и с учетом перекрывания и характера сцепления между последовательностями ДНК-фрагментов. Полноразмерная последовательность исходного ПЦР-продукта может быть получена путем сопоставления с соответствующими экзонами HLA-A/B/С/DQB1 в стандартной базе данных и последующего HLA-A/B/С/DQB1-генотипирования.
1. Экстракция образцов
ДНК экстрагировали из 950 проб крови с использованием известных результатов HLA-SBT-типирования (по Программе забора костного мозга у доноров в Китае, далее называемой CMDP) на автоматическом оборудовании для экстракции KingFisher (US Thermo Co.). Этот метод описан в примере 1.
2. ПЦР-амплификация
950 ДНК, полученных после проведения каждой стадии экстракции образцов, были обозначены No.1-950 и были подразделены на 10 групп (95 ДНК в каждой группе), которые были обозначены HLA-1, HLA-2, HLA-3, HLA-4, HLA-5, HLA-6, HLA-7, HLA-8, HLA-9, HLA-10. Для каждой группы образцов 95 образцов ДНК амплифицировали с использованием 95 наборов ПЦР-праймеров, несущих двунаправленные праймерные индексы (таблица 6) для амплификации экзонов 2, 3, 4 HLA-A/B (таблица 2), для амплификации экзонов 2, 3, 4 HLA-С (таблица 4) и для амплификации экзонов 2, 3 HLA-DQB1 (таблица 5), соответственно. ПЦР-реакцию проводили в 96-луночных планшетах всего в 100 планшетах, обозначенных HLA-X-Р-А2, HLA-X-Р-А3, HLA-X-P-A4, HLA-Х-Р-В2, HLA-X-P-B3, HLA-X-P-B4, HLA-X-P-C2, HLA-X-P-C3, HLA-X-P-С4 и HLA-X-P-DQB1 («X» указывает на номер группы 1/2/3/4/5/6/7/8/9/10, а «А2/3/4», «В2/3/4», «С2/3/4», «DQB1» представляют собой сайты амплификации), где в каждом планшете имеется негативный контроль без какой-либо матрицы, а праймеры, используемые для негативного контроля, представляют собой праймеры, помеченные PI-1 (таблица 6). Во время эксперимента регистрировали информацию о каждом образце по номеру группы и праймерным индексам. Так, например, ниже представлена соответствующая информация о праймерных индексах PI-1 и PI-2, а остальная информация может быть получена аналогичным способом.
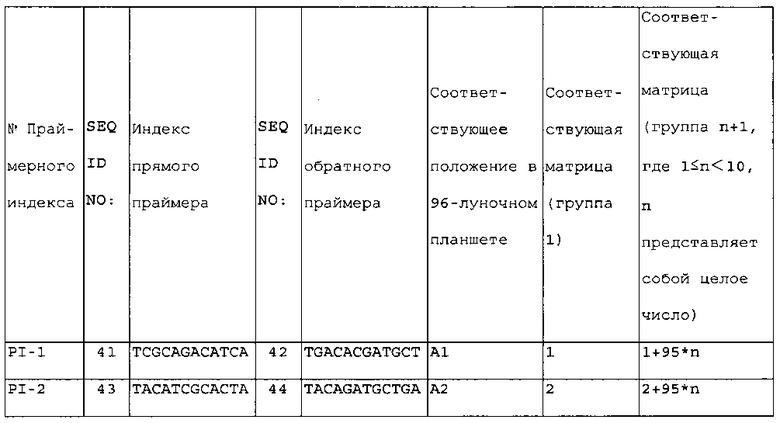
Процедуру ПЦР и систему ПЦР-реакции для HLA-A/B/C осуществляли методом, описанным в примере 2. ПЦР-праймеры для амплификации соответствующих экзонов HLA-A/B представлены в таблице 2, а ПЦР-праймеры для амплификации соответствующих экзонов HLA-C представлены в таблице 4.
ПЦР для HLA-DQB1 проводили в следующих условиях:
96°C 2 мин
95°C 30 сек → 55°C 30 сек → 72°C 20 сек (32 цикла)
15°C ∞
Множество ПЦР-реакций для HLA-DQB1 (одновременной амплификации экзонов 2, 3) проводили с использованием системы, описанной в примере 10, а ПЦР-праймеры для амплификации соответствующих экзонов HLA-DQB1 представлены в таблице 5.
Если PInf-A/B/C-F2/3/4 и PInf-Q-F2/F3 представляют собой F-праймеры HLA-A/B/C/DQB1, имеющие последовательности индекса прямого праймера No. n (таблица 6) у 5'-конца, то PInr-A/B/C-R2/3/4 и PInr-Q-R2/R3 представляют собой R-праймеры HLA-A/B/C/DQB1, имеющие последовательность индекса обратного праймера No. n у 5'-конца (в данном случае n≤95), а остальные праймеры могут быть получены аналогичным образом. Кроме того, каждый образец соответствует конкретной серии ПЦР-праймеров (PInf-A/B/C-F2/3/4, PInr-A/B/C-R2/3/4, PInf-Q-F2/F3, PInr-Q-R2/R3).
ПЦР-реакцию осуществляли на оборудовании для ПЦР PTC-200, поставляемом Bio-Rad Co. После проведения ПЦР 3 мкл ПЦР-продуктов подвергали электрофорезу в 2% агарозном геле. На фигуре 16 представлены результаты электрофореза ПЦР-продуктов соответствующих экзонов HLA-A/B/C/DQB1 образца No.1, а маркером молекулярных масс ДНК является DL 2000 (Takara Co.). На электрофореграмме наблюдалась серия одиночных полос длиной в пределах от 300 п.н. до 500 п.н., что указывало на успешную ПЦР-амплификацию экзонов (A2, A3, A4, B2, B3, B4, C2, C3, C4, DRB1) HLA-A/B/C/DQB1 образца No.1. При этом в негативном контроле (N) полоса амплификации не наблюдалась. Результаты для других образцов были аналогичными.
3. Объединение в пул и очистка ПЦР-продуктов
Для образцов группы X («X» представляет собой 1/2/3/4/5/6/7/8/9/10), 20 мкл остальных ПЦР-продуктов, взятых из каждой лунки 96-луночного планшета с HLA-Х-P-A2 (за исключением негативного контроля), смешивали со встряхиванием в 3-миллилитровой EP-пробирке до получения гомогенной смеси (обозначенной HLA-Х-A2-Mix). Аналогичную процедуру проводили в других девяти 96-луночных планшетах с образцами группы Х, обозначенных HLA-X-A3-Mix, HLA-X-A4-Mix, HLA-X-B2-Mix, HLA-X-B3-Mix, HLA-X-B4-Mix, HLA-X-C2-Mix, HLA-X-C3-Mix, HLA-X-C4-Mix и HLA-X-DQB1-Mix. Из каждого планшета брали 200 мкл смеси HLA-X-A2-Mix, HLA-X-A3-Mix, HLA-X-A4-Mix, HLA-X-B2-Mix, HLA-X-B3-Mix, HLA-X-B4-Mix, HLA-X-C2-Mix, HLA-X-C3-Mix, HLA-X-C4-Mix и HLA-X-DQB1-Mix и смешивали в 3-миллилитровой EP-пробирке, обозначенной HLA-Х-Mix. 500 мкл ДНК-смеси от HLA-Х-Mix подвергали очистке на колонке с использованием набора для очистки ДНК Qiagen (QIAGEN Co.) (Для подробного описания стадий очистки см. инструкции производителей) с получением 200 мкл очищенной ДНК, концентрацию которой определяли на оборудовании Nanodrop 8000 (Thermo Fisher Scientific Co.). Ту же самую процедуру проводили и для других 9 групп образцов. Конечные определенные концентрации ДНК для 10 групп образцов указаны ниже.
Mix
Mix
4. Конструирование библиотек для секвенирования Illumina GA
Как описано в примере 4, ДНК в общем количестве 5 мкг, взятых из очищенной HLA-X-Mix, подвергали фрагментации, а после фрагментации очищали, подвергали реакции репарации концов, присоединяли А к 3'-концу и лигировали с адаптером PCR-FREE-библиотеки Illumina GA.
Ниже показана соответствующая взаимосвязь между группами образцов и адаптерами библиотеки.
Полученные продукты реакции очищали на сферах Ampure (Beckman Coulter Genomics) и растворяли в 50 мкл деионизованной воды, а затем определяли концентрации ДНК с помощью количественной ПЦР с активацией флуоресценции (QPCR) следующим образом:
5. Выделение путем разрезания геля
HLA-1-Mix, HLA-2-Mix, HLA-3-Mix, HLA-4-Mix, HLA-5-Mix, HLA-6-Mix, HLA-7-Mix, HLA-8-Mix, HLA-9-Mix и HLA-10-Mix смешивали в равных молярных концентрациях (конечная концентрация составляла 70,86 нМ/мкл) и обозначали HLA-Mix-10. 30 мкл HLA-Mix-10 подвергали электрофорезу в 2% агарозном геле с низкой температурой плавления. Электрофорез проводили при 100 В, в течение 100 минут. ДНК-маркер представлял собой 50 п.н. ДНК-маркера от NEB Co. Гель, содержащий ДНК-фрагменты размером в пределах от 450 до 750 п.н., разрезали (фигура 17). Продукты, выделенные из разрезанного геля, очищали с помощью набора для ПЦР-очистки QIAquick (QIAGEN Co.), и после такой очистки объем составлял 32 мкл, а концентрация ДНК, измеренная с помощью количественной ПЦР с активацией флуоресценции (QPCR), составляла 10,25 нМ.
6. Секвенирование методом Illumina GA и анализ результатов
Секвенирование и анализ результатов осуществляли методами, описанными в примерах 5 и 6.
Были созданы базы данных, содержащих результаты секвенирования ПЦР-продуктов различных экзонов HLA-A/B/C/DQB1 для каждого образца, соответствующего определенному праймерному индексу. Полученную последовательность ДНК сопоставляли путем выравнивания с последовательностями базы данных для соответствующего экзона HLA-A/B/C/DQB1, имеющегося в профессиональной базе данных HLA IMGT. Если результат выравнивания последовательностей дает 100% соответствие, то это означает, что HLA-A/B/C/DQB1-генотип соответствующего образца был точно определен. См. программу с захватом изображения на экране для конструирования консенсусной последовательности экзона 2 HLA-A-сайта образца No.1, как показано на фигуре 18. Для всех 950 образцов результаты типирования, полученные вышеописанным методом, полностью соответствовали первоначальным известным результатам типирования, где результаты для образцов No.1-32 представлены ниже:


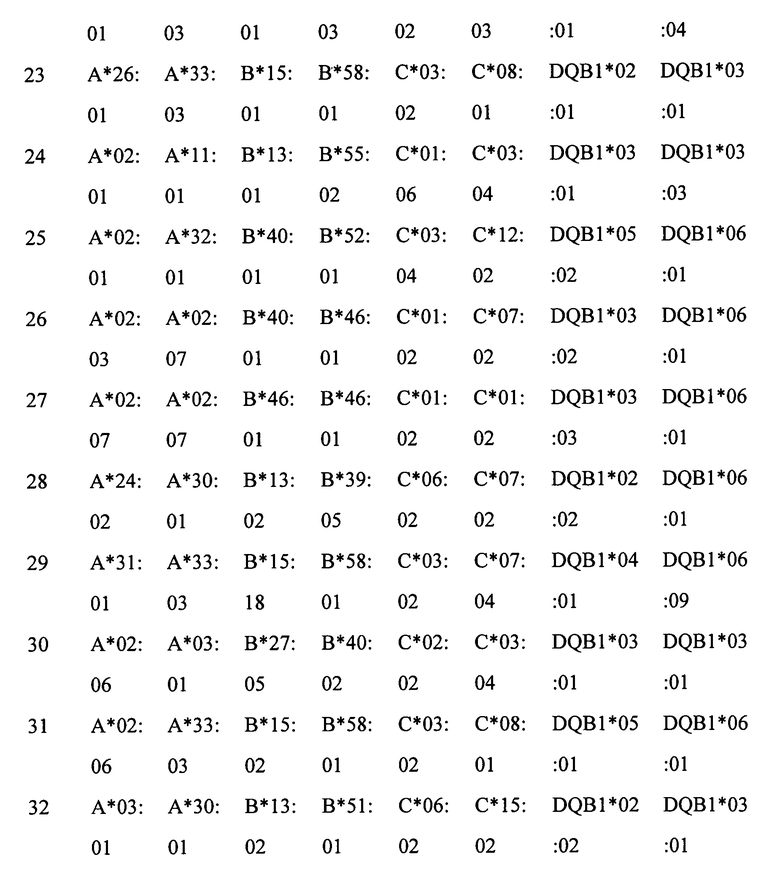

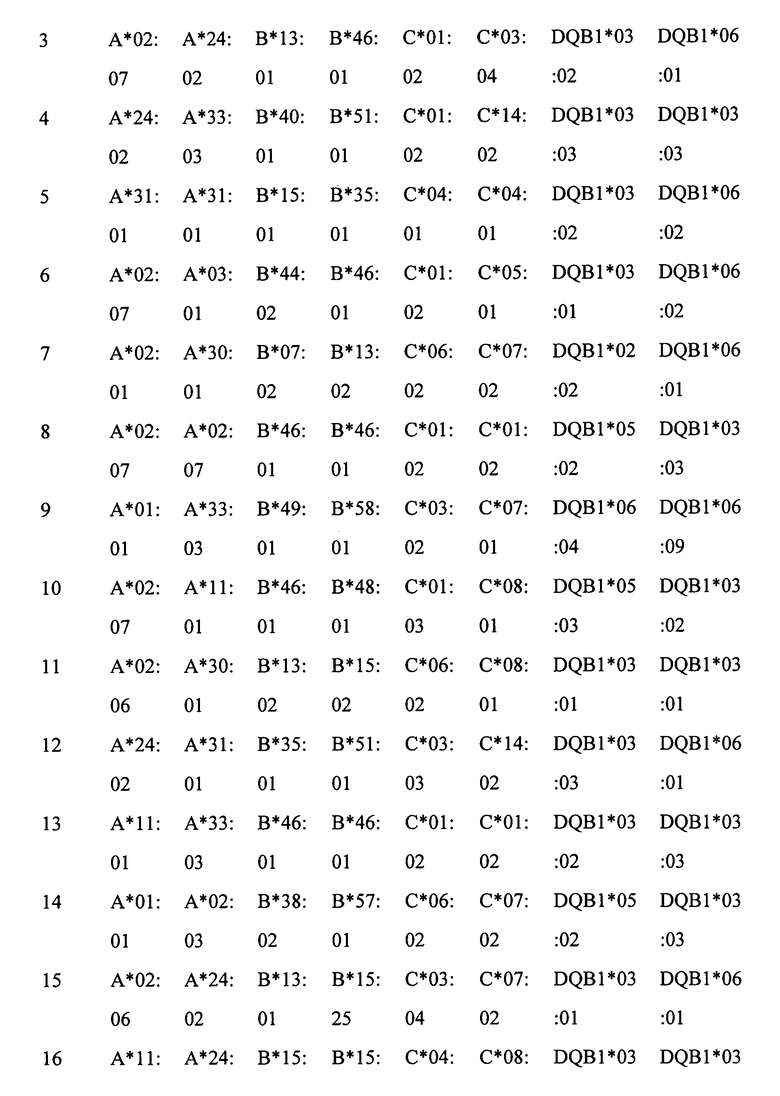
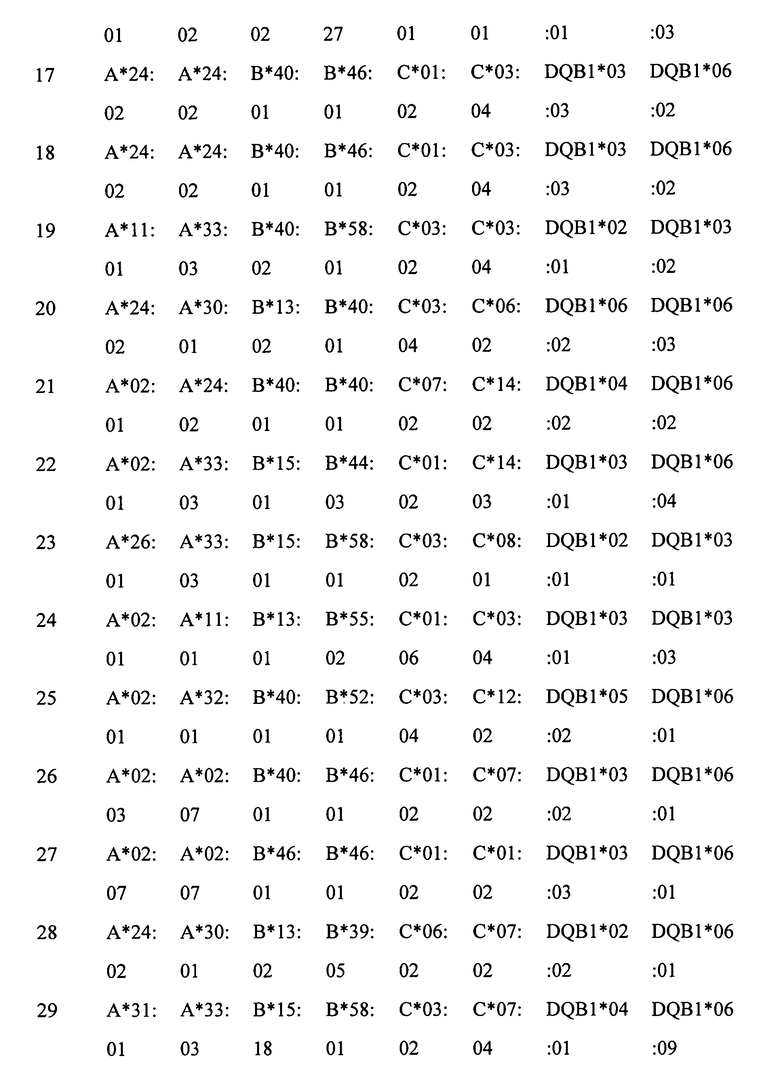
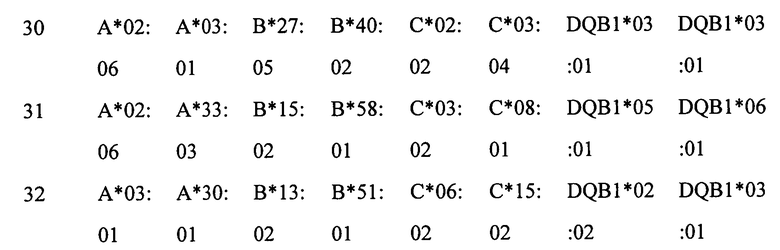
Примечание: В случае если последовательности экзонов 2, 3, 4 HLA-A/B/C были полностью идентичны, то был выбран общий тип.
950 образцов с известными результатами HLA-SBT-типирования подвергали генотипированию в HLA-A/B/C/DQB1-сайтах с применением технической стратегии согласно изобретению, и полученные результаты показали, что данные, полученные с применением технической стратегии согласно изобретению, полностью соответствуют ранее полученным известным данным.
Хотя в настоящей заявке подробно описаны варианты осуществления изобретения, однако для специалиста в данной области очевидно, что исходя из описания изобретения, в него могут быть внесены различные модификации и изменения, не выходящие за рамки существа и объема изобретения. Объем настоящего изобретения определен прилагаемой формулой изобретения и любым ее эквивалентом.
Источники информации
[1]. http://www.ebi.ac.uk/imgt/hla/stats.html.
[2]. Tiercy J M. Molecular basis of HLA polymorphism: implications in clinical transplantation. [J]. Transpl Immunol, 2002, 9:173-180.
[3]. C.Antoine, S.Müller, A.Cant, et al. Long-term survival and transplantation of haemopoietic stem cells for immunodeficiencies: report of the European experience. 1968-99. [J]. The Lancet, 2003, 9357:553-560.
[4]. H. A. Erlich, G. Opelz, J. Hansen, et al. HLA DNA Typing and Transplantation.[J]. Immunity, 2001, 14:347-356.
[5]. Lillo R, Balas A, Vicario JL, et al. Two new HLA class allele, DPB1*02014, by sequence-based typing. [J]. Tissue Antigens, 2002, 59:47-48.
[6]. A. Dormoy, N. Froelich. Leisenbach, et al. Mono-allelic amplification of exons 2-4 using allele group-specific primers for sequence-based typing (SBT) of the HLA-A, -B and -C genes: Preparation and validation of ready-to-use pre-SBT mini-kits. [J]. Tissue Antigens, 2003, 62:201-216.
[7]. Elaine R. Mardis. The impact of next-generation sequencing technology on genetics. [J]. Trends in Genetics. 2008, 24:133-141.
[8]. Christian Hoffmann1, Nana Minkah1, Jeremy Leipzig. DNA barcoding and pyrosequencing to identify rare HIV drug resistance mutations. [J]. Nucleic Acids Research, 2007, 1-8.
[9]. Shannon J. Odelberg, Robert B. Weiss, Akira Hata. Template-switching during DNA synthesis by Thermus aquaticus DNA polymerase I. [J]. Nucleic Acids Research. 1995, 23:2049-2057.
[10]. Sayer D, Whidborne R, Brestovac B. HLA-DRB1 DNA sequencing based typing: an approach suitable for high throughput typing including unrelated bone marrow registry donors. [J]. Tissue Antigens. 2001, 57(1):46-54.
[11]. Iwanka Kozarewa, Zemin Ning, Michael A Quail. Amplification-free Illumina sequencing-library preparation facilitates improved mapping and assembly of (G+C)-biased genomes. [J]. Nature Methods. 2009, 6:291-295.
[12]. Marsh, S.G.E., Parham, P. & Barber, L.D. The HLA Facts Book 3-91 (Academic Press, London, 2000).
[13]. Campbell, K.J. et al. Characterization of 47 MHC class I sequences in Filipino cynomolgus macaques. Immunogenetics 61, 177-187 (2009).
[14]. Goulder, P.J.R. & Watkins, D.I. Impact of MHC class I diversity on immune control of immunodeficiency virus replication. Nat. Rev. Immunol. 8, 619-630 (2008).
[15]. O'Leary, C.E. et al. Identification of novel MHC class I sequences in pig-tailed macaques by amplicon pyrosequencing and full-length cDNA cloning and sequencing. Immunogenetics 61, 689-701 (2009).
[16]. Robinson J, Malik A, Parham P, Bodmer JG, Marsh SGE.IMGT/HLA database-a sequence database for the human major histocompatibility complex. Tissue Antigens 55, 80-7 (2000).
[17]. Hoffmann C, Minkah N, Leipzig J, Wang G, Arens MQ, Tebas P, Bushman FD. DNA bar coding and pyrosequencing to identify rare HIV drug resistance mutations. Nucleic Acids Res. 2007; 35(13):e91.
[18]. WU, D. L. et al. Comparative analysis of serologic typing and HLA-II typing by micro-PCR-SSP. Di Yi Jun Yi Da Xue Xue Bao, 2002, 22:247-249.
[19]. Al- Hussein K A, Rama N R, Butt A I, et al. HLA class II sequence based typing in normal Saudi individuals. Tissue Antigens, 2002, 60: 259-261.
[20]. D. C. Sayer, D. M. Goodridge. Pilot study: assessment of interlaboratory variability of sequencing-based typing DNA sequence data quality. Tissue Antigens, 2007, 69 Suppl: 66-68.
[21]. Horton V, Stratton I, Bottazzo G. F. et al. Genetic heterogeneity of autoimmune diabetes: age of presentation in adults is influenced by HLA DRB1 and DQB1 genotypes. Diabetologia, 1999, 42:608-616.
[22]. C.E.M. Voorter, M.C. Kik1, E.M. van den Berg-Loonen et al. High-resolution HLA typing for the DQB1 gene by sequence-based typing. Tissue Antigens, 2008, 51:80-87.
[23]. G. Bentley, R. Higuchi, B. Hoglund et al. High-resolution, high-throughput HLA genotyping by next-generation sequencing. Tissue Antigens, 2009, 74:393-403.
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ пробоподготовки образцов для типирования генов главного комплекса гистосовместимости HLA-A, HLA-B, HLA-C, HLA-DPB1, HLA-DQB1, HLA-DRB1 и олигонуклеотидные праймеры для его реализации | 2023 |
|
RU2829344C1 |
| Способ секвенирования экзонов гена HLA-DQA1 и набор синтетических олигонуклеотидов для его реализации | 2024 |
|
RU2833819C1 |
| Способ секвенирования экзонов гена HLA-DPA1 и набор синтетических олигонуклеотидов для его реализации | 2024 |
|
RU2837865C1 |
| НАБОР СИНТЕТИЧЕСКИХ ОЛИГОНУКЛЕОТИДОВ ДЛЯ ОПРЕДЕЛЕНИЯ ПОСЛЕДОВАТЕЛЬНОСТИ ГЕНОВ HLA-A, HLA-B, HLA-C, HLA-DQB1, HLA-DRB1 ГЛАВНОГО КОМПЛЕКСА ГИСТОСОВМЕСТИМОСТИ | 2019 |
|
RU2703545C1 |
| ПРЯМОЙ ЗАХВАТ, АМПЛИФИКАЦИЯ И СЕКВЕНИРОВАНИЕ ДНК-МИШЕНИ С ИСПОЛЬЗОВАНИЕМ ИММОБИЛИЗИРОВАННЫХ ПРАЙМЕРОВ | 2011 |
|
RU2565550C2 |
| СПОСОБ ОБНАРУЖЕНИЯ МИКРОДЕЛЕЦИЙ В ОБЛАСТИ ХРОМОСОМЫ С ДНК-МАРКИРУЮЩИМ УЧАСТКОМ | 2012 |
|
RU2610691C2 |
| ТРАНСПОЗИЦИЯ С СОХРАНЕНИЕМ СЦЕПЛЕНИЯ ГЕНОВ | 2015 |
|
RU2709655C2 |
| СЛОЖНЫЕ КОМПЛЕКСЫ СВЯЗАННОЙ НА ПОВЕРХНОСТИ ТРАНСПОСОМЫ | 2020 |
|
RU2790295C2 |
| Способ получения молекулярных STR-маркеров Х-хромосомы для идентификации неизвестного индивида и определения биологического родства для работы с образцами малых количеств ДНК и набор олигонуклеотидов для его осуществления | 2021 |
|
RU2800084C2 |
| Способ получения молекулярных маркеров для определения вероятной внешности человека методом мультиплексной амплификации для работы с образцами деградированной ДНК | 2021 |
|
RU2786246C2 |

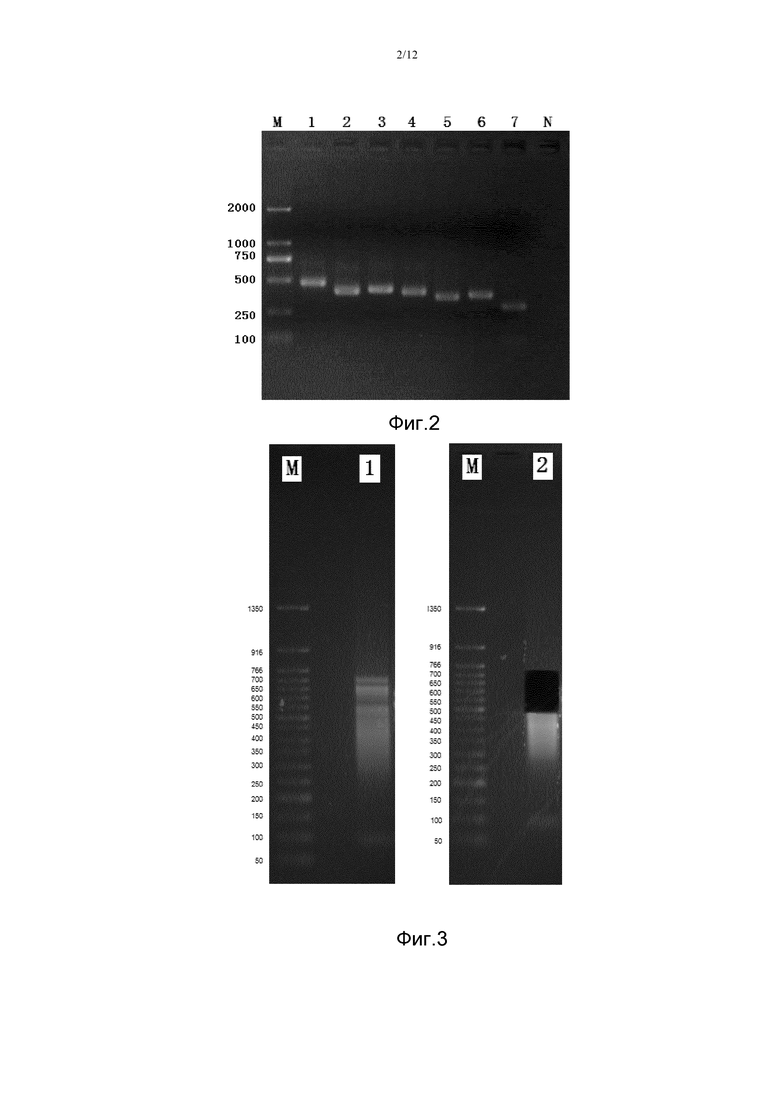

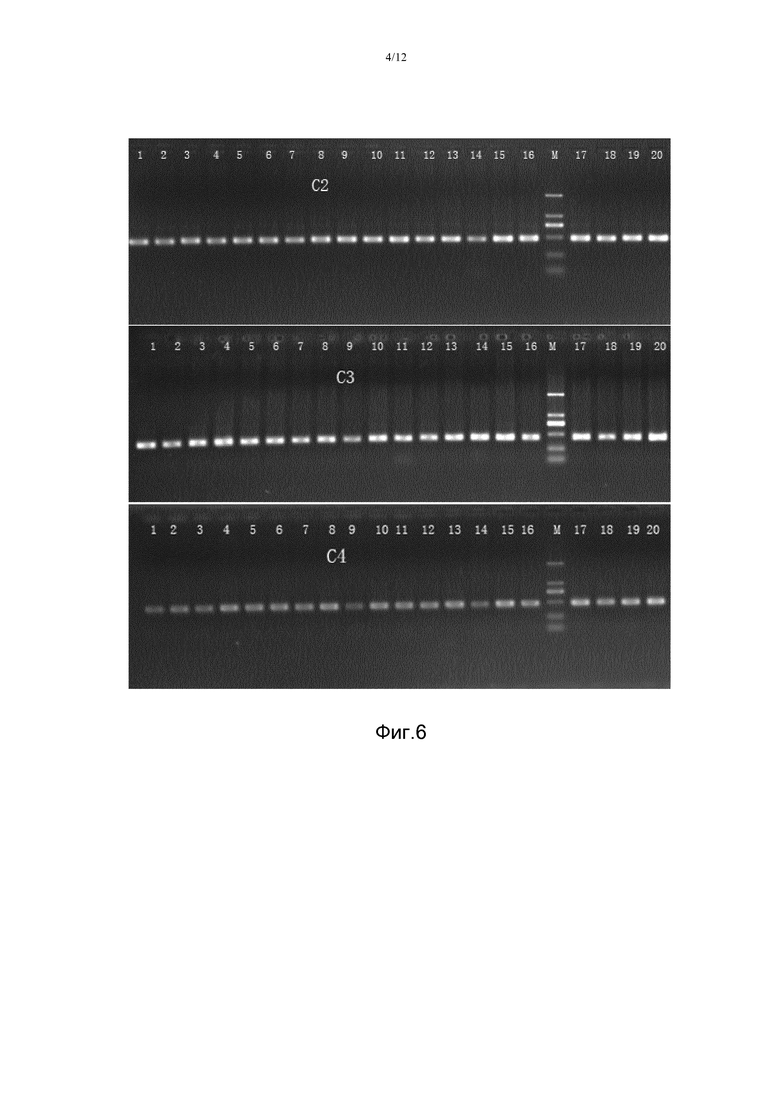


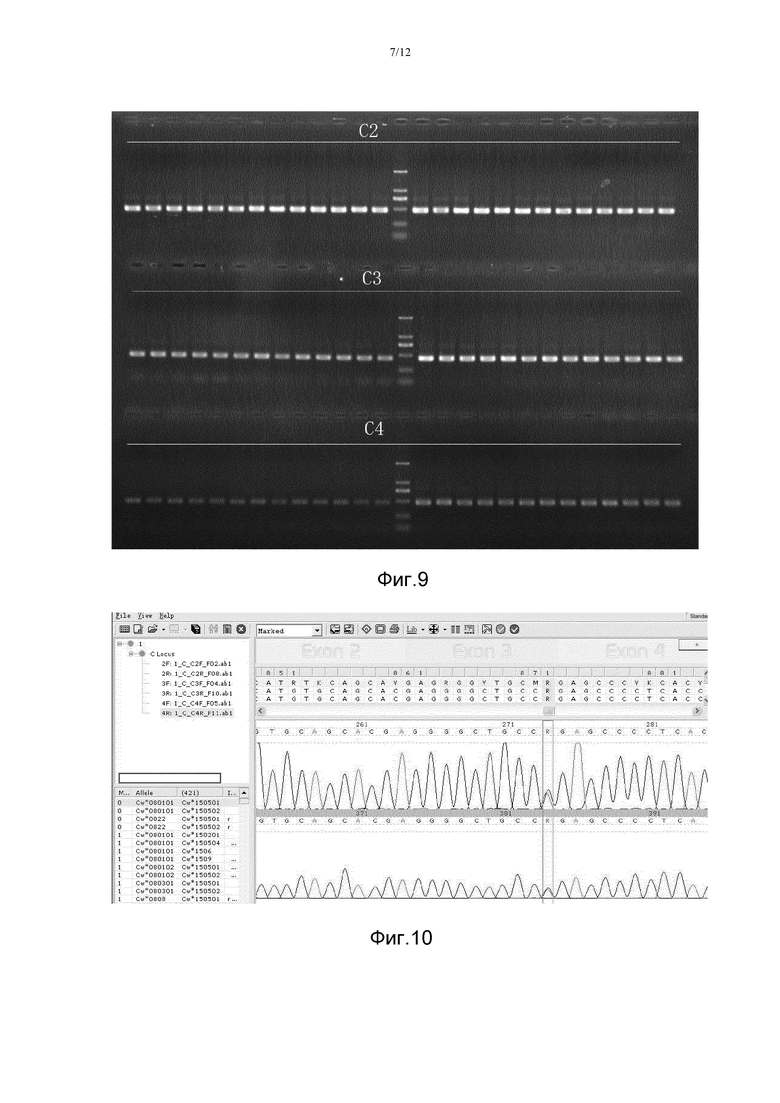
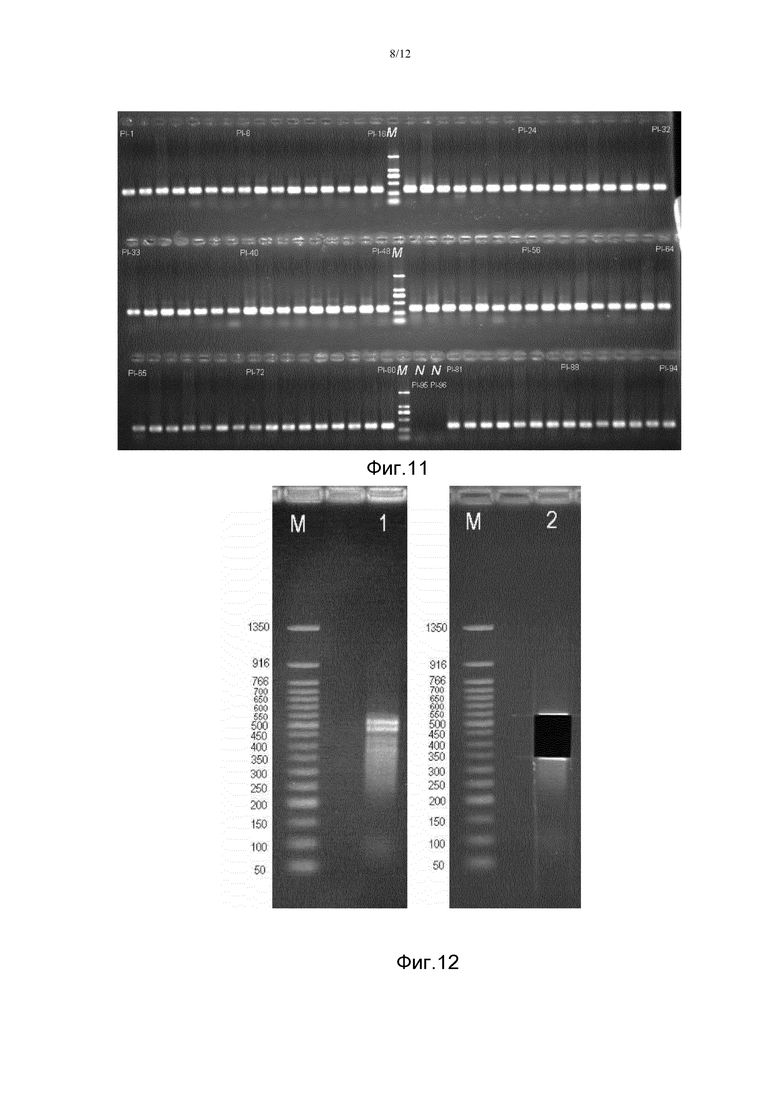

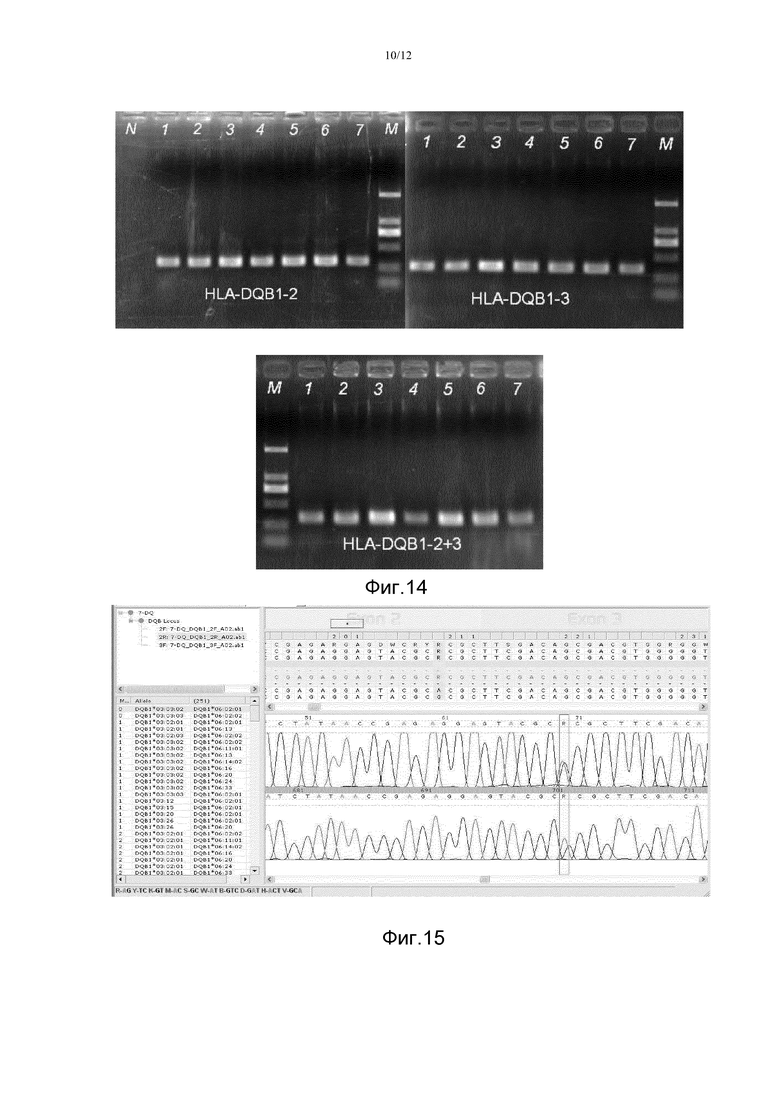
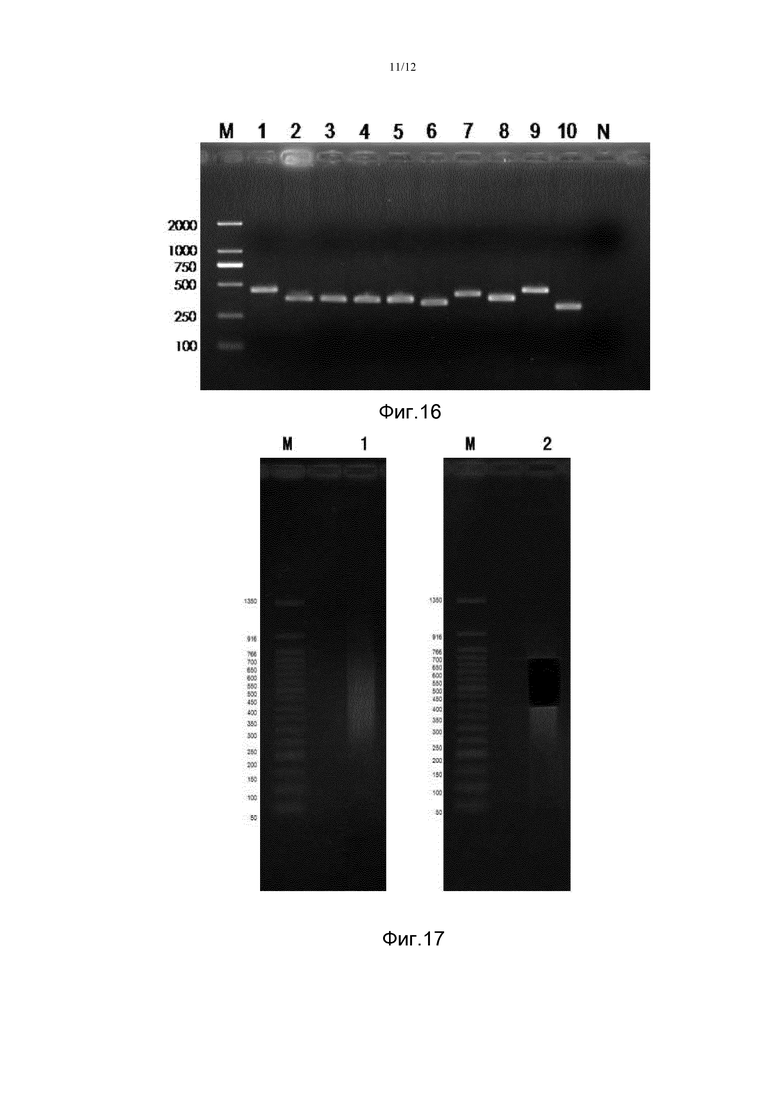
















































Изобретение относится к биохимии. Описан способ ПЦР-секвенирования, включающий следующие стадии: получение образца; амплификация; смешивание; фрагментация; секвенирование и сплайсинг. Настоящее изобретение также относится к праймерным меткам, используемым в указанном способе, а также к способу генотипирования, в частности, в анализе HLA. Настоящее изобретение также относится к ПЦР-праймерам для HLA-A/B, HLA-C и HLA-DQB1. Изобретение позволяет повысить длину продукта ПЦР, который может быть секвенирован секвенатором, без повышения цены и/или редукции. 3 н. и 16 з.п. ф-лы, 18 ил., 13 табл., 12 пр.
1. Способ определения нуклеотидной последовательности представляющей интерес нуклеиновой кислоты в образце, где указанный способ включает:
1) получение n образцов, где n представляет собой целое число ≥1; причем необязательно указанные n образцов, предназначенные для анализа, разделяют на m групп, где m представляет собой целое число и n≥m≥1;
2) амплификацию, где пару или множество пар индексированных праймеров используют для каждого образца, если от этого образца имеются матрицы; ПЦР-амплификацию осуществляют в условиях, подходящих для амплификации представляющей интерес нуклеиновой кислоты, где каждая пара индексированных праймеров состоит из прямого индексированного праймера и обратного индексированного праймера, содержащих праймерные индексы, где праймерные индексы, присутствующие в прямом индексированном праймере и в обратном индексированном праймере каждой пары индексированных праймеров, могут быть одинаковыми или различными и где индексы праймеров в парах индексированных праймеров, используемых для различных образцов, являются различными;
где каждая пара индексированных праймеров образована парой праймерных индексов и парой ПЦР-прймеров, где прямой и обратный ПЦР-праймеры имеют прямой праймерный индекс и обратный праймерный индекс, необязательно связанные линкерной последовательностью на 5′-конце, соответственно;
3) объединение в пул, где если n>1, то ПЦР-продукты от каждого образца объединяют в один пул;
4) фрагментирование, где амплифицированные продукты подвергают неполному фрагментированию, очистке и выделению;
5) секвенирование, где выделенную ДНК-смесь подвергают секвенированию методом секвенирования второго поколения с получением последовательностей фрагментированной ДНК; и
6) сборку, где устанавливают соответствие между полученными данными секвенирования и образцами один за другим по уникальному праймерному индексу для каждого образца; риды каждой последовательности сопоставляют путем выравнивания с эталонной последовательностью ДНК, соответствующей ПЦР-продуктам, с помощью программы выравнивания; сборку полноразмерной последовательности представляющей интерес нуклеиновой кислоты осуществляют из последовательностей фрагментированной ДНК по перекрыванию и характеру сцепления последовательностей.
2. Способ по п. 1, где указанная пара праймерных индексов выбрана из 95 пар праймерных индексов, показанных в Таблице 6; и указанные ПЦР-праймеры выбраны из SEQ ID NO: 1-40 и 231-238.
3. Способ по п. 1 или 2, где указанные ПЦР-праймеры выбраны из:
(1) ПЦР-праймеров для амплификации экзонов 2, 3 и 4 HLA-A/B, как показано в таблице 1 или в таблице 2;
(2) ПЦР-праймеров для амплификации экзона 2 HLA-DRB1, как показано в таблице 7;
(3) ПЦР-праймеров для амплификации экзонов 2, 3 и/или 4 HLA-С, представленных в таблице 3 или в таблице 4; и
(4) ПЦР-праймеров для амплификации экзона 2 и/или 3 гена HLA-DQB1, представленных в таблице 5.
4. Способ по п. 1 или 2, где указанные праймерные индексы содержат по меньшей мере 10, или по меньшей мере 20, или по меньшей мере 30, или по меньшей мере 40, или по меньшей мере 50, или по меньшей мере 60, или по меньшей мере 70, или по меньшей мере 80, или по меньшей мере 90, или все 95 пар из 95 пар праймерных индексов, представленных в таблице 6; и/или
где указанные праймерные индексы состоят из 10-95 пар, 20-95 пар, 30-95 пар, 40-95 пар, 50-95 пар, 60-95 пар, 70-95 пар, 80-95 пар, 90-95 пар или 95 пар из 95 пар праймерных индексов, представленных в таблице 6; и/или
где указанные праймерные индексы включают по меньшей мере PI-1-PI-10, или PI-11-PI-20, или PI-21-PI-30, или PI-31-PI-40, или PI-41-PI-50, или PI-51-PI-60, или PI-61-PI-70, или PI-71-PI-80, или PI-81-PI-90, или PI-91-PI-95 из 95 пар праймерных индексов, представленных в таблице 6, или комбинации любых двух или более из указанных пар.
5. Способ по п. 1 или 2, где указанное фрагментирование ДНК включает методы химического фрагментирования и методы физического фрагментирования, где методы химического фрагментирования включают ферментативное расщепление, а методы физического фрагментирования включают методы ультразвукового фрагментирования или методы механического фрагментирования.
6. Способ по п. 1 или 2, где после указанного фрагментирования ДНК все ДНК-полосы, имеющие длину между максимальной длиной ридов в секвенаторе и максимально приемлемой длиной ДНК в секвенаторе, очищают и выделяют, где указанные методы очистки и выделения включают выделение путем электрофореза и разрезания геля и выделение на магнитных сферах.
7. Способ по п. 1, где указанный способ, помимо стадий 1)-4) п. 1, включает следующие стадии:
5) конструирование библиотеки, где конструирование PCR-FREE-библиотеки для секвенирования осуществляют с использованием библиотеки фрагментированных ПЦР-продуктов, где различные адаптеры библиотеки могут быть добавлены для различения различных PCR-FREE-библиотек для секвенирования и где все ДНК-полосы, имеющие длину между максимальной длиной ридов в секвенаторе и максимально приемлемой длиной ДНК в секвенаторе, очищают и выделяют;
6) секвенирование, где выделенную ДНК-смесь подвергают секвенированию методом секвенирования второго поколения с получением последовательностей фрагментированных ДНК; и
7) сборку, где устанавливают соответствие между полученными данными секвенирования и образцами один за другим по различным последовательностям адаптеров библиотеки для библиотек и по уникальному праймерному индексу для каждого образца; каждую последовательность рида сопоставляют путем выравнивания с эталонной последовательностью ДНК, соответствующей ПЦР-продуктам, с помощью программы выравнивания; а сборку полноразмерной последовательности представляющей интерес нуклеиновой кислоты осуществляют из последовательностей фрагментированной ДНК по перекрыванию и характеру сцепления последовательностей.
8. Способ по п. 7, где на стадии 5) очищают и выделяют фрагменты ДНК длиной 450-750 п.о.
9. Способ по п. 1 или 7, где прямой индексированный праймер и/или обратный индексированный праймер представляют собой вырожденные праймеры и/или где метод секвенирования второго поколения представляет собой парно-концевой метод.
10. Способ по п. 1 или 7, где метод секвенирования второго поколения представляет собой метод секвенирования Illumina GA или Illumina Hiseq 2000.
11. Способ по п. 1 или 7, где программа выравнивания представляет собой Blast или BWA.
12. Способ по п. 1 или 2, где указанные образцы берут у млекопитающего.
13. Способ по п. 12, где указанные образцы берут у человека.
14. Способ по п. 12, где указанные образцы представляет собой пробы человеческой крови.
15. Применение способа по любому из пп. 1-14 в HLA-типировании, где указанный способ отличается тем, что он включает секвенирование взятого у пациента образца указанным способом по любому из пп. 1-13 и сопоставление результатов секвенирования путём выравнивания с данными о последовательностях экзонов 2, 3, 4 HLA-A/B, экзонов 2, 3 и/или 4 HLA-C, экзона 2 и/или 3 гена HLA-DQB1 и/или экзона 2 HLA-DRB1 в базе данных HLA; где если результат выравнивания последовательностей дает 100% соответствие, то это означает, что HLA-генотип соответствующего образца был точно определен.
16. Применение по п. 15, где указанный образец представляет собой пробу крови и/или где указанная база данных HLA представляет собой профессиональную базу данных HLA IMGT.
17. Способ по п. 1 или 2, где указанный способ используется для способа HLA-типирования и дополнительно включает следующую стадию:
типирование, где результаты секвенирования сопоставляют с данными о последовательностях экзонов 2, 3, 4 HLA-A/B, экзонов 2, 3 и/или 4 HLA-C, экзона 2 и/или 3 гена HLA-DQB1 и/или экзона 2 HLA-DRB1 в базе данных HLA; где если результат выравнивания последовательностей дает 100% соответствие, то это означает, что HLA-генотип соответствующего образца был точно определен.
18. Способ по п. 17, где указанная база данных HLA представляет собой профессиональную базу данных HLA IMGT.
19. Применение набора в способе по любому из пп. 1-14, где указанный набор включает пару или совокупность пар индексированных праймеров и, необязательно, средства для амплификации ДНК, очистки ДНК и/или секвенирования ДНК, где каждая пара индексированных праймеров включает пару праймерных индексов и пару ПЦР-прймеров, где прямой и обратный ПЦР-праймеры имеют прямой и обратный праймерные индексы, необязательно связанные линкерной последовательностью на 5′-конце, соответственно; где указанная пара праймерных индексов выбрана из 95 пар праймерных индексов, показанных в Таблице 6; и указанные ПЦР-праймеры выбраны из SEQ ID NO: 1-40 и 231-238.
| US2009170713 A1, 02.07.2009 | |||
| WO2007140540 A2, 13.12.2007 | |||
| WO2005042764 A2, 12.05.2005 | |||
| US7300755 B1, 27.11.2007 | |||
| WO9835059 A1, 13.08.1998 | |||
| JEESUK YU ET AL., Analysis of children with type 1 diabetes in Korea: high prevalence of specific anti-islet autoantibodies, immunogenetic similarities to Western populations with "unique" haplotypes, and lack |
Авторы
Даты
2016-06-20—Публикация
2011-06-30—Подача