Область техники, к которой относится изобретение
Изобретение относится к области молекулярной биологии, биотехнологии, судебной медицины и криминалистики, генетическим исследованиям в области идентификации человека.
Уровень техники
В настоящее время анализ ДНК-профилей индивидов является наиболее доказательным методом анализа биологического материала при производстве судебно-медицинской идентификационной экспертизы и используется в криминалистике с 1985 года. ДНК-анализ позволяет исследовать специфичные для каждого человека участки генома, и получить, таким образом, уникальный генетический "паспорт" или генетически профиль индивида. Индивидуализирующие признаки, определяемые на уровне ДНК, характеризуются почти абсолютной устойчивостью, то есть сохраняются в организме человека неизменными на продолжении всей жизни. В последние десятилетия для целей генетической идентификации личности широко применяются различные маркерные системы, локализованных как на аутосомах, так и на нерекомбинирующих участках Y-хромосомы и Х-хромосом и в митохондриальной ДНК.
В начале 1990-х годов короткие тандемные повторы (STR) были введены в качестве нового типа полиморфного ДНК-маркера и с тех пор стали золотым стандартом идентификации человека в базах данных ДНК, уголовных дел, анализа отцовства и родства, а также идентификации пропавших без вести лиц. Успехи в разработке больших мультиплексных STR-панелей значительно увеличили силу дискриминации, что помогает в принятии и исключении решений об идентификации человека.
Аутосомные STR и STR Y-хромосомы (Y-STR) широко применяются в судебно-медицинском анализе ДНК в течение многих лет. Однако в последние годы STR-значения Х-хромосомы (X-STR) стали рассматриваться как важные инструменты для судебно-медицинской экспертизы и анализа родства [1]. Уникальная структура Х-хромосомы, сформированная эволюцией, привела к нынешним гендерно-специфическим генетическим различиям. У мужчин рекомбинация между Х- и Y-хромосомами ограничена псевдоавтосомными областями; следовательно, у мужчин Х-хромосома (почти) полностью передается потомству женского пола. С другой стороны, Х-хромосома присутствует у женщин в двух копиях, которые рекомбинируют по всей хромосоме во время мейоза и передаются потомкам как женского, так и мужского пола. Эти характеристики передачи, помимо очевидного клинического воздействия (анеуплоидии половых хромосом очень часты), делают Х-хромосому незаменимым генетическим инструментом для популяционных генетических исследований, а также для исследований родства и судебно-медицинской экспертизы.
Специфические свойства Х-хромосомы - две рекомбинирующие копии у женщин и одна нерекомбинирующая копия у мужчин (за исключением областей PAR), создающие гаплотипы - обеспечивают маркерам Х-хромосомы особое место в криминалистике и популяционной генетике, а также в других областях исследований, таких как эволюционные исследования человека и медицинская генетика (например, Х-сцепленные рецессивные расстройства, такие как гемофилия или мышечная дистрофия Дюшенна).
Использование полиморфизмов Х-хромосомы для задач идентификации человека и в популяционной генетике в основном обуславливается их уникальными свойствами. Исключая или дополняя информацию, предоставляемую аутосомными маркерами или маркерами, расположенными на Y-хромосоме или мтДНК, маркеры Х-хромосомы могут предоставить важную информацию во многих различных направлениях исследований. В стандартных наборах для идентификации обычно используется аутосомные STR-маркеры, в то время как доступные в настоящее время маркеры Х-хромосомы в некоторых случаях могут обеспечить большую статистическую достоверность [2]. Следует подчеркнуть, что тестирование личности с использованием X-STR в определенных контекстах, а именно в сценариях (сложного) тестирования родства, может быть единственным инструментом для раскрытия определенных случаев [3]. Среди уже присутствующих на рынке зарубежных наборов, можно отметить набор Investigator® Argus Х-12 QS (компания QIagen) для мультиплексной амплификации, состоящий из 12 STR-локусов X-хромосомы, амелогенина и D21S11.: DXS10074, DXS10079, DXS10101, DXS10103, DXS10134, DXS10135, DXS10146, DXS10148, DXS71326 DXS7423, DXS8378, HPRTB), который использует метод фрагментного анализа.
Набор Forenseq, который содержит 7 Х- STR- маркеров (DXS10074, DXS10103, DXS10135, DXS7132, DXS7423, DXS8378, HPRTB), используется метод "массового параллельного секвенирования" ( DNA Signature Prep Kit (Verogen, San Diego, CA, USA).
DNA Signature Prep Kit (Verogen, San Diego, CA, USA).
Имеющиеся в настоящее время разработки зарубежных наборы, содержащие панель STR-маркеров для Х-хромосомы не всегда адаптированы для криминалистических образцов плохого качества, где содержится малое количество ДНК. Кроме этого, существенным недостатком импортных наборов для ДНК-идентификации является их значительная дороговизна, обусловленная необходимостью использования дорогостоящих расходных материалов (специфических ДНК-полимераз, растворов для ПЦР, буферных растворов). Для устранения указанных недостатков необходимым условием является повышение информативности предлагаемого набора и его экономичности, а также адаптации для специфики используемых в криминалистических экспертизах образцов биологического материала, где ДНК содержится в малом количествах.
Наиболее часто распространенные 12 маркеров Х-хромосомы, которые используются в зарубежных наборах для идентификации, сгруппированы в четыре группы сцепления (по три маркера на группу), и, таким образом, каждый набор из трех маркеров обрабатывается как гаплотип для генотипирования при использовании стандартного метода КЭ (капилярного электофореза). Группы сцепления маркеров выглядят следующим образом:
(Xp11) - DXS7132 - DXS10074 - DXS10079,
(Хр22) DXS8378 - DS10135 - DXS10148,
(Хр26) - HPRTB - DXS10101 - DXS10103,
(Хр28) - DXS7423 - DXS10134 - DXS10146
Вышеописанные 12 STR-маркеров послужили авторам изобретения основой для разработки собственного набора маркеров. При этом авторами были учтены некоторые моменты, ограничивающие использование только данных 12 маркеров применительно к методу "массового параллельного секвенирования" (NGS-секвенирование), а именно - размер повторяющейся последовательности для маркеров DXS10146 и DXS10134 (Хр28) и DXS10101 (Хр26) превышает размер 200 п.о., и, следовательно, размер ПЦР-продуктов (ампликонов) будет больше 250 п.о. Для некоторых часто встречаемых аллельных состояний такой размер ПЦР-продуктов мог превышать 400 п.о. При использовании метода одноконцевого "массового параллельного секвенирования" для определения нуклеотидной последовательности ампликона длина продукта этих маркеров является слишком большой, в связи с чем корректное генотипирование таких маркеров будет затруднительно. Поэтому, в разработанный в данном изобретении оригинальный набор маркеров авторами были добавлены дополнительные маркеры меньшего размера их тех же областей X хромосомы. Выбраны были следующие дополнительные маркеры: DXS10075, DXS8377 и DXS10147, к которым были подобраны олигонуклеотидные праймеры.
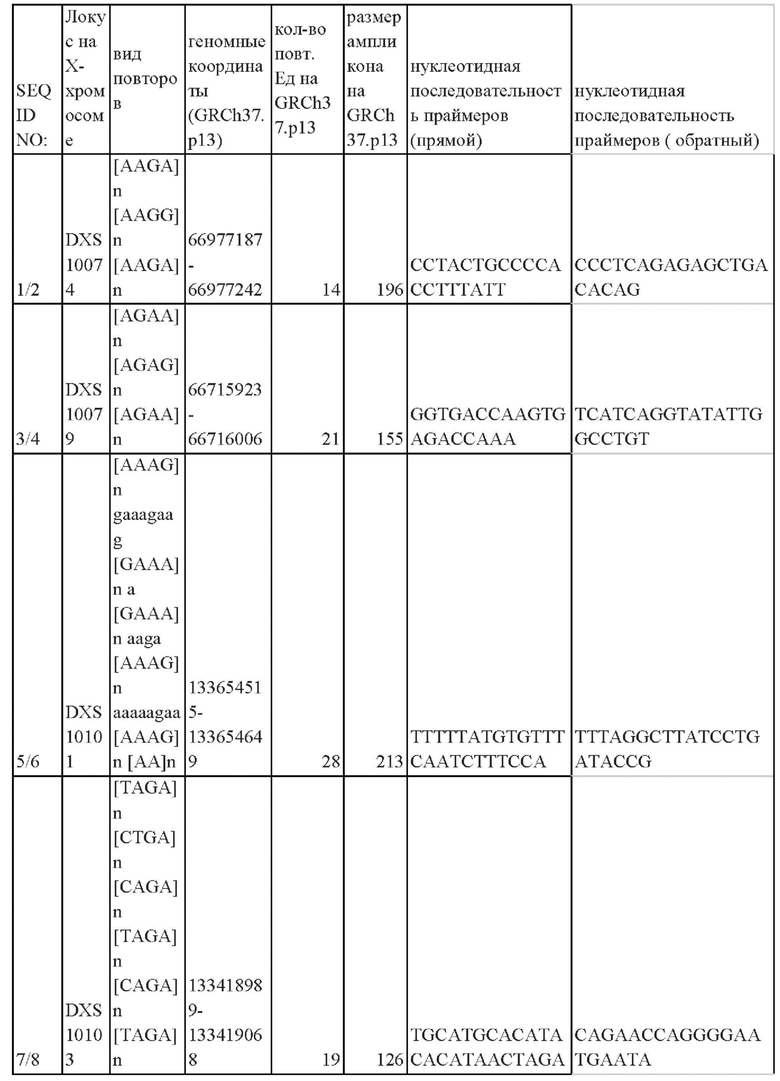
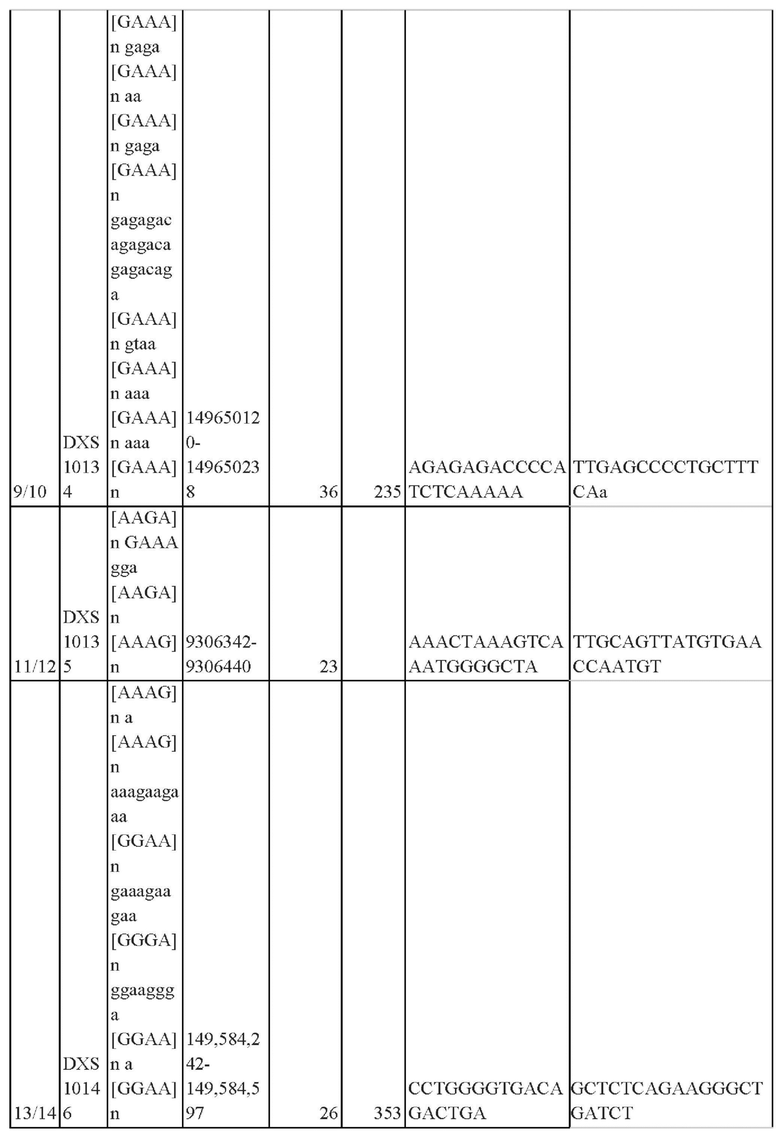
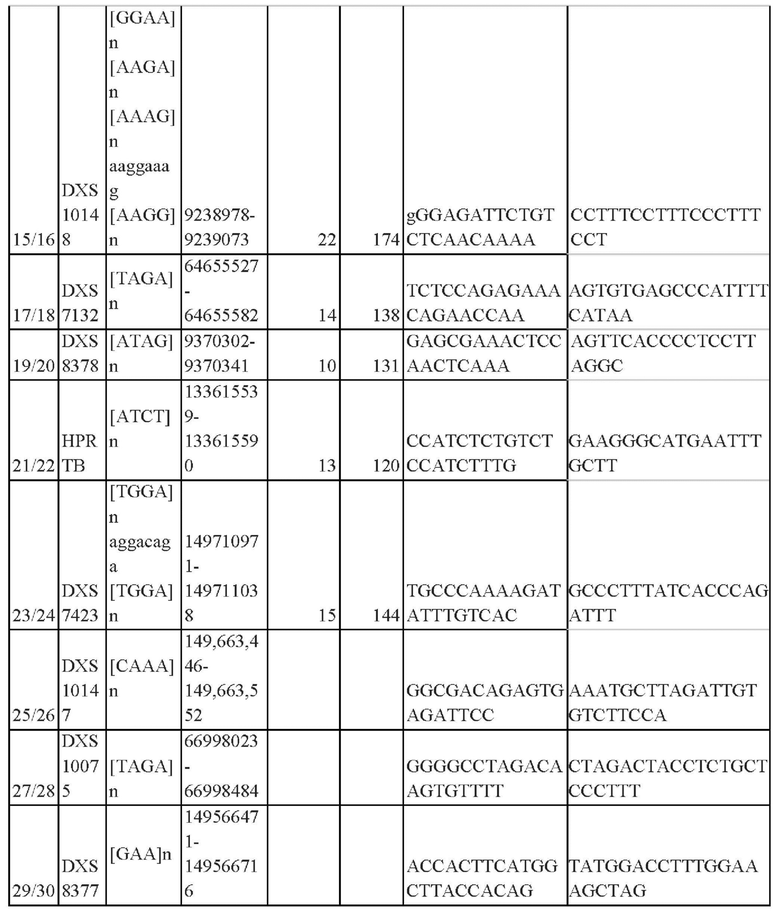
Таким образом, авторами был предложен набор из 15 пар синтетических олигонуклеотидных праймеров для амплификации локусов, содержащих STR-маркеры X-хромосомы человека (DXS7423, DXS10148, DXS10159, DXS6809, DXS7424, DXS8378, DXS1079164, DXS101, DXS10103, DXS10101, HPTRB, DXS10075, DXS10074, DXS10135 и DXS10134), адаптированный для анализа образцов, в которых ДНК находится в малом количестве методом "массового параллельного секвенирования". Набор для генотипирования включает композицию, образованную 15 парами синтетическими оригинальными олигонуклеотидными праймерами для локусов в геноме человека, включающих STR-повторы на Х-хромосоме, позволяющую проводить мультиплексную ПЦР (нуклеотидная последовательность праймеров SEQ ID NO: 1- SEQ ID NO 30 приведена в таблице 1).
Преимущества данного набора состоит в том, что авторами изобретения создана система олигонуклеотидов, позволяющая одновременно анализировать множество образцов методом "массового параллельного секвенирования", и данная система олигонуклеотидов адаптирована для использования на малых количествах биологического материала (ДНК).

Осуществление способа
Пример генотипирования биологического образца
В примере показан способ получения выбранных 15 STR-маркеров Х-хромосомы человека на биологических образцах, содержащих малые количества ДНК человека, путем проведения мультиплексного ПЦР-анализа с последующим масштабным параллельным секвенированием полученных мультиплексных ПЦР-продуктов и генотипированием выбранных маркеров с использованием адаптированной программы STRin NGS [4].
Получение ДНК
ДНК может быть получена из любого биологического материала человека, любой ткани человека, содержащей клеточные ядра, например, лейкоцитов, волосяных фолликулов, клеток буккального эпителия и др. Пригодная для проведения ПЦР ДНК может быть выделена из образа при помощи любого ранее широко применяемого метода выделения ДНК. Авторы рекомендуют использовать готовые набороы для выделения ДНК из различных тканей человека (например, представленных фирмой Qiagen)
Получение молекулярных STR-маркеров Х-хромосомы включает в себя четыре этапа:
- Мультиплексная ПЦР геномной ДНК с использованием смеси специфических олигонуклеотидных праймеров;
- Приготовление фрагментных библиотек из полученных ПЦР-продуктов для секвенирования на платформе Illumina;
- Проведение широкомасштабного параллельного секвенирования полученных библиотек.
Амплификация участков генома, содержащих исследуемые маркеры
Амплификация участков генома, содержащих исследуемые маркеры, может быть осуществлена с помощью наборов Multiplex PCR kit компании «Qiagen», на приборах GeneAmp PCR System 9700 Thermal Cycler компании «Applied Biosystems».
Состав реакционной смеси для мультиплексной ПЦР:
- Раствор олигонуклеотидов для мультиплексной ПЦР (в конечной концентрации от 1 нМ до 0,5 мкМ
- Смесь полимеразы и буфера
- Деионизированная вода
- Оптимизирующий раствор
- Исследуемая ДНК.
Набор олигонуклеотидных праймеров адаптирован для использования ДНК в количестве от 1 нг до 100 пг ДНК. При необходимости образец ДНК следует развести в буфере ЕВ. Для проведения работ с набором реагентов для ДНК-идентификации неизвестного индивида по деградированной ДНК и ДНК из малых количеств биоматериала рекомендуется использовать. Минимальная концентрация препарата ДНК - 4,2 пг/мкл.
Также необходимо проводить параллельно контрольную реакцию (отрицательный контроль), где вместо ДНК добавляется деионизированная вода.
Амплификацию следует проводить в пробирках, стрипах, или планшетах предназначенных для ПЦР.
В отдельной стерильной пробирке смешать из расчета на одну реакцию все необходимые компоненты, согласно расчетной таблице (пробирка с раствором MasterMix).
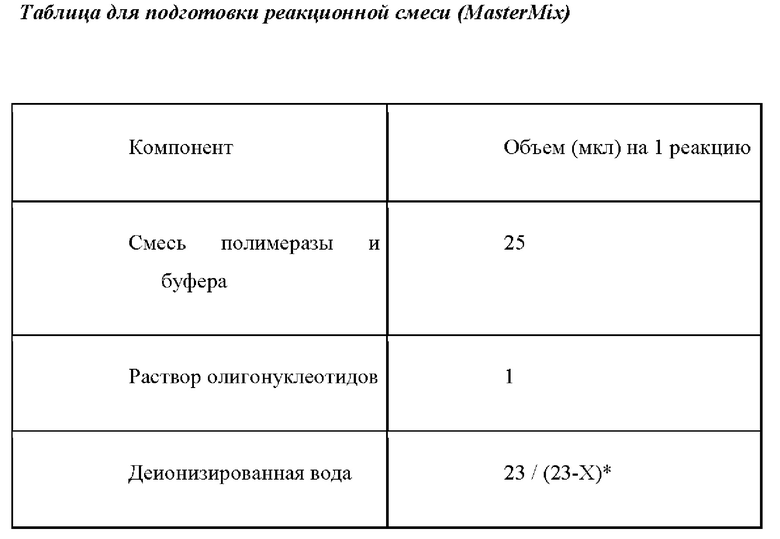
При использовании такой системы для амплификации (2-х смесь буфера и полимеразы) минимальная возможная для использования концентрация ДНК составляет 4,2 пг/мкл.
После приготовления смесь для амплификации помещают в амплификатор и выставляется программа:
95°С - 15 минут 94°С - 30 секунд
57°С - 90 секунд 35 циклов
72°С - 90 секунд 72°С - 10 минут
4°С∝.
Наличие продуктов амплификации исследуемой ДНК и отсутствие продуктов амплификации в отрицательном контроле проверяют с помощью электрофореза в агарозном геле. Продукты амплификации очищают от остаточных праймеров с помощью соответствующих мини-колонок (например, Qiagen MinElute system) в соответствии с инструкцией к используемому набору реагентов. Полученные в результате мультиплексной амплификации очищенные ПЦР продукты, содержащие маркеры для идентификации неизвестного индивида используются в качестве матрицы для приготовления фрагментных геномных библиотек для секвенирования.
Приготовление фрагментных библиотек
Предварительно необходимо провести измерение концентрации очищенных ПЦР-фрагментов, полученных на предыдущем этапе мультиплексной амплификации на приборах NanoDrop или Qubit (ThermoFisher) в соответствии с инструкцией к прибору. Для приготовления фрагментных библиотек оптимально использовать около 300 нг ДНК очищенных ПЦР-продуктов мультиплексной амплификации. Приготовление фрагментных геномных библиотек проводят с использованием наборов реагентов Illumina®. TruSeq® DNA PCR-Free library prep kit в соответствии с инструкцией к набору. Для секвенирования нескольких образцов фрагментных библиотек одновременно необходимо использовать индексированные адаптеры (например, Dual index adaptors Illumina). Для каждого образца библиотеки используется отдельный индексируемый адаптер, который необходимо записать в таблицу для дальнейшего учета при анализе результатов секвенирования нескольких образцов фрагментных библиотек. Для приготовления фрагментных библиотек рекомендуется/возможно использовать 1/2 от объема всех реагентов, входящих в набор Illumina®. TruSeq® DNA PCR-Free library prep kit, прописанного в инструкции к набору.
Этапы 1-3 являются рекомендованными, но не обязательными.
1. Очистка фрагментных библиотек: необходимо нанести весь объем элюата в одну лунку 2% агарозного геля с красителем SYBR Gold (например, с использованием готового 2%-ного E-gel (Invitrogen)). Провести электрофорез с использованием маркера молекулярных весов 50 bp Ladder в течение 10 минут (в случае использования системы e-gel (hrvitrigen)).
2. С использованием стерильного одноразового скальпеля вырезать кусочки геля, с фрагментами библиотеки от 220 п.н. и больше. Светящиеся фрагменты длиной меньше 150 п.н. соответствуют димерам адаптеров и не должны быть использованы для дальнейшего анализа.
3. Провести очистку вырезанных фрагментов ДНК с использованием набора реагентов MinElute gele extraction kit (Qiagen). Эллюировать в 20-22 мкл буфера ЕВ (Qiagen).
Приготовленные описанным выше способом фрагментные библиотеки можно напрямую использовать для секвенирования на технологической платформе Illumina, предварительно проведя оценку качества полученных фрагментных библиотек.
Качественную и количественную оценку фрагментных библиотек можно проводить стандартными методами, рекомендованными фирмой производителем платформ для секвенирования Illumina. Для оптимизации рабочего процесса рекомендуем использовать следующие этапы: измерить концентрации фрагментных библиотек на приборе Qubit с использованием набора реагентов HS. Для дальнейшего секвенирования рекомендуется использовать фрагментные библиотеки с концентрацией не менее 2 нМ/мкл.
Перед секвенированием необходимо провести мультиплексирование библиотек в эквимолярных количествах (объединение библиотек для нескольких образцов). Не допускается объединение в один пул библиотек с одинаковыми индексами.
Приготовленные фрагментные библиотеки секвенируют методом параллельного широкомасштабного секвенирования на технологической платформе Illumina в режиме одноконцевого секвенирования с длинной прочтения не менее 350 п.о. Рекомендуемый прибор секвенатор MiSeq (Illumina). Определение генотипов проводили с помощью адаптированной программы STRin NGS [4].
Авторами были проанализированы 20 образцов ДНК индивидов разного пола (мужчин и женщин) в различных разведениях (1 нг, 500 пкг, 100 пкг, 25 пкг) и контрольная ДНК М2800(Promega). В результате работы, у всех исследованных образцов с разным разведением ДНК были амплифицированные все выбранные локусы. Однако, при использовании ДНК в разведении равном 25 мкг, около 20% локусов либо не амплифицировались (не наблюдалось ПЦР-продуктов этих локусов), либо и наблюдалось выпадение отдельных аллелей, следует отметить, что этот результат был характерен только для исследованных образцов женского пола.
Список литературы
1. Iva Gomes et al. Twenty Years Later: A Comprehensive Review of the X Chromosome Use in Forensic GeneticsFront Genet. 2020 Sep 17; 11:926
2. Szibor et al., 2003; Krawczak, 2007; Szibor, 2007; Pinto et al., 2011a, 2013a; Gomes et al., 2012)
3. Pinto N., Gusmão L., Amorim A. (2011), Pinto N., Gusmão L., Egeland L.Т., Amorim A. (2013) Paternity exclusion power: comparative behaviour of autosomal and X-chromosomal markers in standard and deficient cases with inbreeding. Forensic Sci. Int. Genet. 7 290-295.). X-chromosome markers in kinship testing: a generalisation of the IBD approach identifying situations where their contribution is crucial. Forensic Sci. Int. Genet. 5 27-32.
4. Carina Grøntved Jønck, Xiaoqin Qian, Halimureti Simayijiang, Claus Børsting, STRinNGS v2.0: Improved tool for analysis and reporting of STR sequencing data, Forensic Science International: Genetics, Volume 48, 2020, 102331, ISSN 1872-4973, https://doi.org/10.1016/j.fsigen.2020.102331.
Способ получения молекулярных STR-маркеров Х-хромосомы методом мультиплексной амплификации для работы с образцами малых количеств ДНК.
Перечень последовательностей синтетических оригинальных олигонуклеотидных праймеров SEQ ID NO: 1- SEQ ID NO: 30
SEQ ID NO 1
CCTACTGCCCCACCTTTATT
SEQ ID NO 2
CCCTCAGAGAGCTGACACAG
SEQ ID NO 3
GGTGACCAAGTGAGACCAAA
SEQ ID NO 4
TCATCAGGTATATTGGCCTGT
SEQ ID NO 5
TTTTTATGTGTTTCAATCTTTCCA
SEQ ID NO 6
TTTAGGCTTATCCTGATACCG
SEQ ID NO 7
TGCATGCACATACACATAACTAGA
SEQ ID NO 8
CAGAACCAGGGGAATGAATA
SEQ ID NO 9
AGAGAGACCCCATCTCAAAAA
SEQ ID NO 10
TTGAGCCCCTGCTTTCAА
SEQ ID NO 11
AAACTAAAGTCAAATGGGGCTA
SEQ ID NO 12
TTGCAGTTATGTGAACCAATGT
SEQ ID NO 13
CCTGGGGTGACAGACTGA
SEQ ID NO 14
GCTCTCAGAAGGGCTGATCT
SEQ ID NO 15
GGGAGATTCTGTCTCAACAAAA
SEQ ID NO 16
CCTTTCCTTTCCCTTTCCT
SEQ ID NO 17
TCTCCAGAGAAACAGAACCAA
SEQ ID NO 18
AGTGTGAGCCCATTTTCATAA
SEQ ID NO 19
GAGCGAAACTCCAACTCAAA
SEQ ID NO 20
AGTTCACCCCTCCTTAGGC
SEQ ID NO 21
CCATCTCTGTCTCCATCTTTG
SEQ ID NO 22
GAAGGGCATGAATTTGCTT
SEQ ID NO 23
TGCCCAAAAGATATTTGTCAC
SEQ ID NO 24
GCCCTTTATCACCCAGATTT
SEQ ID NO 25
GGCGACAGAGTGAGATTCC
SEQ ID NO 26
AAATGCTTAGATTGTGTCTTCCA
SEQ ID NO 27
GGGGCCTAGACAAGTGTTTT
SEQ ID NO 28
CTAGACTACCTCTGCTCCCTTT
SEQ ID NO 29
ACCACTTCATGGCTTACCACAG
SEQ ID NO 30
TATGGACCTTTGGAAAGCTAG
Изобретение относится к области биотехнологии, судебной медицины и криминалистики, генетическим исследованиям в области идентификации человека. Предложен способ получения молекулярных STR-маркеров Х-хромосомы методом мультиплексной амплификации для идентификации неизвестного индивида и определения биологического родства для работы с образцами малых количеств ДНК и набор синтетических олигонуклеотидных праймеров для проведения мультиплексной полимеразной цепной реакции (ПЦР). Набор для генотипирования включает композицию, образованную 15 парами синтетических оригинальных олигонуклеотидных праймеров для STR-локусов Х-хромосомы человека. Изобретение позволяет успешно анализировать малые количества биологического материала для идентификации неизвестного индивида и определения биологического родства в российской популяции с высокой чувствительностью, специфичностью и точностью. 2 н. и 3 з.п. ф-лы, 2 табл., 1 пр.
1. Способ получения молекулярных STR-маркеров Х-хромосомы методом мультиплексной амплификации для идентификации неизвестного индивида и определения биологического родства для работы с образцами малых количеств ДНК, характеризующийся тем, что для генотипирования используют композицию из 15 пар оригинальных синтетических олигонуклеотидных праймеров для проведения мультиплексной полимеразной цепной реакции (ПЦР) с нуклеотидными последовательностями SEQ ID NO: 1-SEQ ID NO: 30, фланкирующих участки ДНК, содержащие STR-маркеры Х-хромосомы человека; ПЦР-продукты, соответствующие выбранным праймерам, представляют собой маркеры.
2. Способ по п. 1, отличающийся тем, что полученные в результате мультиплексной амплификации ПЦР-продукты, содержащие STR-маркеры Х-хромосомы человека используют в качестве матрицы для фрагментных библиотек ДНК для последующего определения нуклеотидной последовательности продуктов амплификации (ПЦР-продуктов) секвенированием по методу "массового параллельного секвенирования".
3. Способ по п. 1, отличающийся тем, что биологический образец содержит ДНК в малом количестве, менее 1 нг.
4. Способ по п. 1 для идентификации неизвестного индивида, происходящего из популяций Российской Федерации.
5. Набор олигонуклеотидов для получения молекулярных STR-маркеров Х-хромосомы способом по п. 1, используемых для генотипирования индивида, включающий синтетические оригинальные олигонуклеотиды с нуклеотидными последовательностями SEQ ID NO: 1-SEQ ID NO 30.
| ForenSeq™ DNA Signature Prep Kit Achieve high resolution and exceptional accuracy, even with complex mixtures, or degraded DNA, 06.2018, https://verogen.com/wp-content/uploads/2018/08/ForenSeq-prep-kit-data-sheet-VD2018002.pdf | |||
| РЕБРИКОВ Д.В | |||
| и др., ПЦР в реальном времени, 6-е издание, БИНОМ, лаборатория знаний, Москва, 2015, с | |||
| Устройство для выпрямления опрокинувшихся на бок и затонувших у берега судов | 1922 |
|
SU85A1 |
| Основы | |||
Авторы
Даты
2023-07-18—Публикация
2021-11-11—Подача