Изобретение относится к биотехнологии и молекулярной биологии и связано с производством специального вида расходных материалов - маркеров длин фрагментов нуклеиновых кислот. Они могут быть использованы медицинскими диагностическими лабораториями, лабораториями экспертизы и научно-исследовательскими лабораториями при проведении всех видов гель-электрофоретического анализа нуклеиновых кислот. Сущность изобретения заключается в новом способе получения маркерных лестниц, заключающемся в наработке фрагментов ДНК или РНК, составляющих лестницу, с помощью реакции амплификации "катящимся кольцом" (АКК) в варианте рамификации по синтетической кольцевой матрице заданного размера. Предлагаемый способ позволяет получать маркерные лестницы в значительном количестве с любым размерным шагом в большом диапазоне.
Описание чертежей
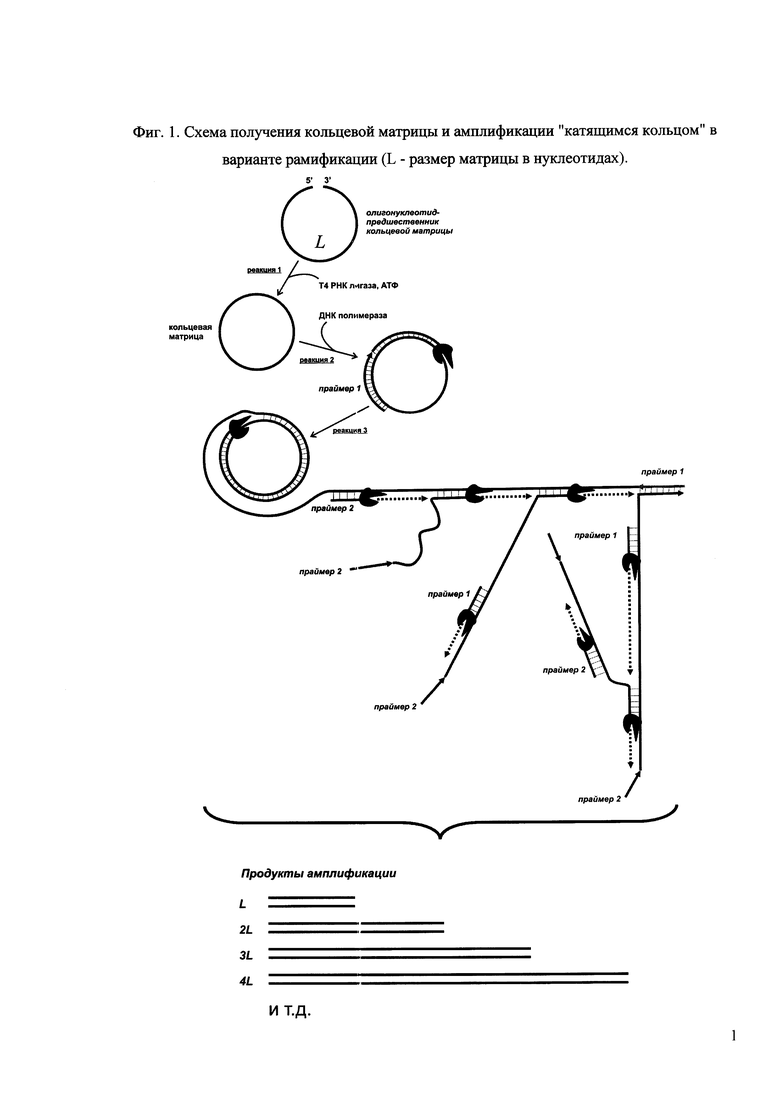
Фиг. 1. Схема получения кольцевой матрицы и амплификации "катящимся кольцом" в варианте рамификации (L - размер матрицы в нуклеотидах).
Фиг. 2. Электрофореграмма маркерных лестниц: М - коммерческий маркер, 1 - маркерная лестница, полученная с помощью кольцевой матрицы размером 50 нуклеотидов, 2 - маркерная лестница, полученная с помощью кольцевой матрицы размером 60 нуклеотидов, 3 - маркерная лестница, полученная с помощью кольцевой матрицы размером 100 нуклеотидов.
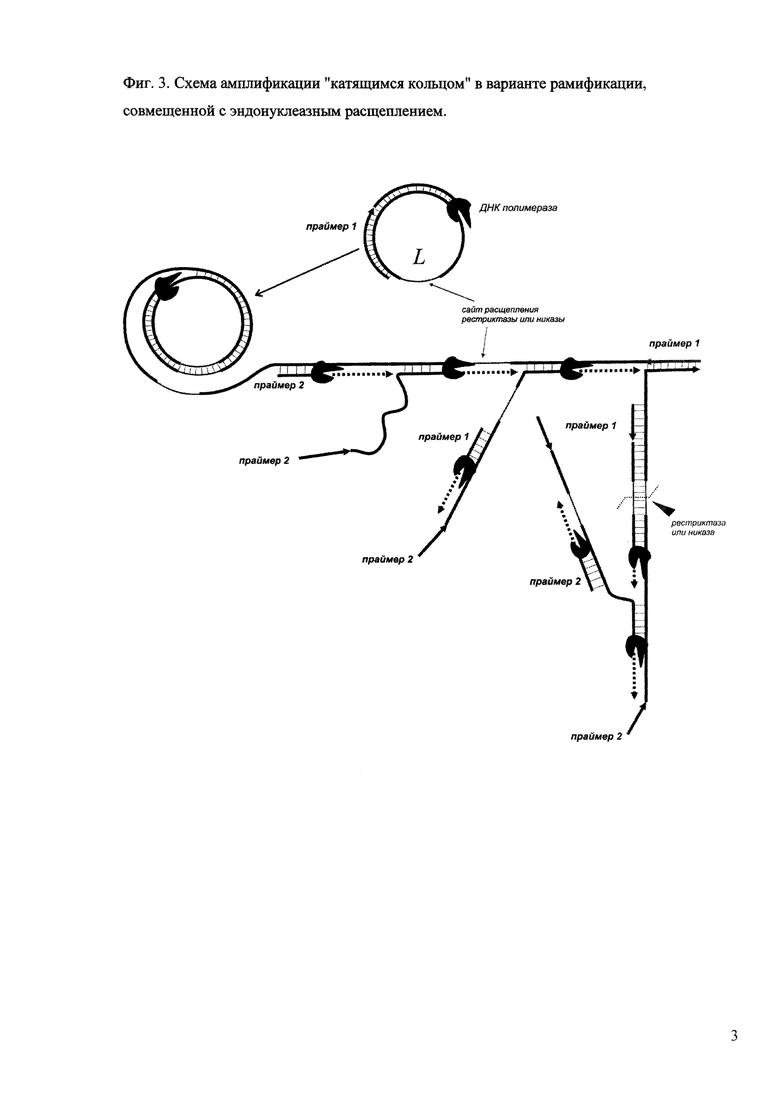
Фиг. 3. Схема амплификации "катящимся кольцом" в варианте рамификации, совмещенной с эндонуклеазным расщеплением.
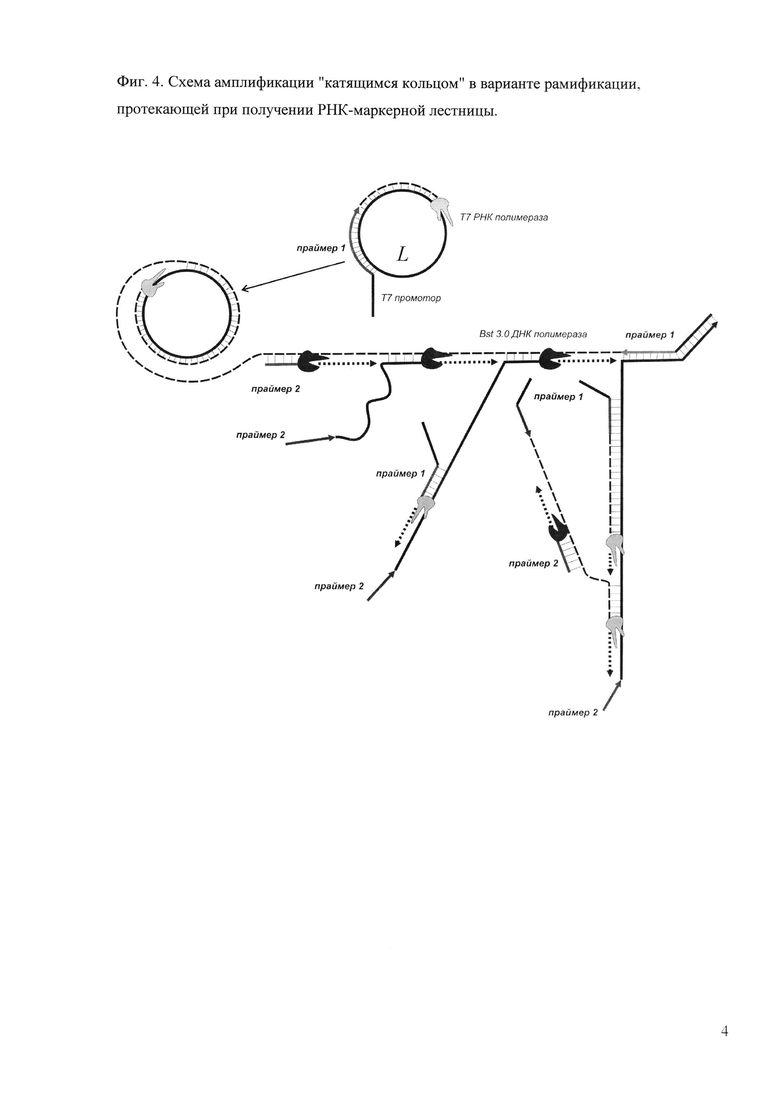
Фиг. 4. Схема амплификации "катящимся кольцом" в варианте рамификации, протекающей при получении РНК-маркерной лестницы.
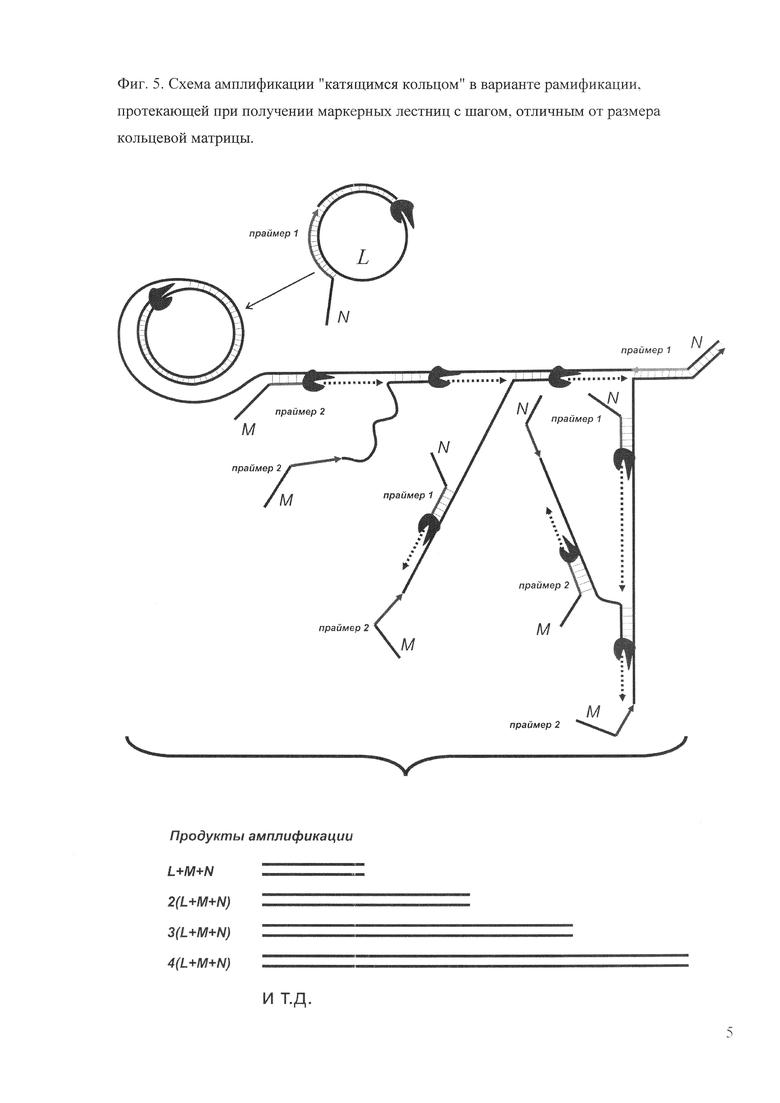
Фиг. 5. Схема амплификации "катящимся кольцом" в варианте рамификации, протекающей при получении маркерных лестниц с шагом, отличным от размера кольцевой матрицы.
Описание изобретения
Обнаружение рестрикционных эндонуклеаз и их использование для расщепления молекул ДНК на фрагменты определенной длины придало импульс электрофоретическому анализу нуклеиновых кислот в полиакриламидных и агарозных гелях. Для сравнения между собой результатов разных электрофоретических разделений потребовалось использование маркерных молекул известной длины, соотнесение с которыми по электрофоретической подвижности позволило более точно определять размеры фрагментов ДНК, образованных под действием рестриктаз. Среди первых маркерных молекул были ДНК фагов и плазмид с определенными размерами рестриктазных фрагментов или с известными нуклеотидными последовательностями. Наиболее широко используемыми были фаг лямбда и плазмида pBR322. До того как стала известна полная последовательность фага лямбда, размер его рестриктазных фрагментов предварительно оценивался с помощью электронной микроскопии [Murray K., Murray N., 1975; Southern, 1979]. Для определения размеров мелких фрагментов использовались химически синтезированные ДНК [Maniatis et al., 1975]. Плазмидный вектор pBR322, широко использовавшийся для молекулярного клонирования, после полного секвенирования [Sutcliffe, 1978; 1979] стал применяться и в качестве маркера молекулярных масс фрагментов ДНК малого размера.
В книге "Gel Electrophoresis on Nucleic Acids", изданной в серии "Practical Approaches in Biochemistry Series", в разделе, посвященном гель-электрофорезу ДНК [Sealey, Southern, 1982], рекомендовалось использовать ДНК тех фагов и плазмид, для которых стали известны нуклеотидные последовательности, чтобы можно было точно рассчитать размеры рестриктазных фрагментов. В Приложении 1 к книге [Minter, Sealey, 1982] были приведены таблицы, в которых для ряда фагов и плазмид указаны размеры фрагментов ДНК, образуемых под действием различных рестрикционных эндонуклеаз с определенными к тому времени нуклеотидными последовательностями. В каталогах ряда фирм в начале 80-х гг.прошлого века стали появляться специальные позиции - продукты расщепления разными рестриктазами (или их смесями) молекул фаговой и плазмидной ДНК, служащие маркерами для гель-электрофореза.
Другим типом маркеров являются маркерные «лестницы» ДНК, «ступеньки» которых одинаковы и относительно равномерно располагаются в гелевом треке, а интенсивность свечения полос разного размера, как правило, сближена. Впервые маркерную лестницу ДНК со ступеньками в 123 п. н. стала поставлять американская фирма Bethesda Research Laboratories, в каталоге которой за 1983 г. в разделе "Gel Markers: Nucleic Acids and Protein" (pp. 22-25) наряду с маркерами из рестриктазных фрагментов ДНК фага лямбда появился инновационный продукт - 123 bp Ladder. Через год эта же фирма вывела на рынок килобазную лестницу.
Начало использованию маркерных лестниц ДНК было положено работой американских авторов [Hartley, Gregori, 1981], в которой они сообщили об успешном клонировании 34 повторов короткого участка гена пролактина крысы. О применении клонированных коротких повторов в качестве маркерных лестниц авторы в статье не упоминают. Однако при взгляде на электрофореграммы в статье возникает идея о возможности такого применения, и именно эта рекомбинантная плазмида стала источником коммерческого продукта 123 bp Ladder [Hartley, Gregori, 1983]. Схожий подход был запатентован в 1998 г. [Hyman, 1998]. Его главным отличием было наличие дополнительного сайта рестриктазы, расположенного на некотором удалении от таких же сайтов, ограничивающих повторы из 100 п.н., что позволило исключить в верхней части геля аморфную полосу, принадлежащую остаткам вектора с прилегающими к нему повторяющими участками. Более сложная плазмида, содержащая несколько кассет из коротких повторов длиной 10 п.н., при выщеплении которых с помощью рестриктазы EcoRV в условиях недорестрикции формировалась 10 п.н. маркерная ДНК-лестница, сконструирована авторами [Hu et al., 2005].
В 2012 г. вьетнамские авторы создали рекомбинантую плазмиду, содержащую 8 повторов по 100 п.н. [Lan et al., 2012]. Отступив с каждого края этой мультивставки на 100 п.н., они подобрали к плазмидным последовательностям праймеры, которые при амплификации давали продукт размером в 1000 п.н. Его неполное расщепление рестриктазой SmaI приводило к появлению лестницы полос со ступеньками в 100 п.н. Воспользовавшись тем, что геномная ДНК мучного хрущака Tenebrio molitor несет высококопийные повторы (десятки миллионов копий), мономерная единица которых с сайтом рестриктазы EcoRI внутри повтора составляет 142 п.н., израильские авторы [Barvish et al., 2007] предложили для создания маркерной лестницы ДНК проводить данным ферментом недорасщепление тотальной ДНК этого насекомого. В результате формировалась характерная лестница полос.
В 2010 г. китайские авторы [Ye et al., 2010] создали иную генно-инженерную конструкцию, в которой вставка, клонированная в несколько приемов, состояла из 15 повторов, но каждый повтор был больше предыдущего на мономерную единицу размером 100 п.н. При расщеплении созданной рекомбинантной плазмиды общим размером 8700 п.н. рестриктазой EcoRI в условиях полной рестрикции образовался набор фрагментов от 100 до 1500 п.н.. Несколько ранее другими китайскими авторами [Dongyi et al., 2008] были созданы две рекомбинантные плазмиды, в которых они клонировали амплифицированные участки фага лямбда. При расщеплении рестриктазой EcoRI созданных в итоге двух плазмид pYO и pYE образовывались маркерные лестницы со ступеньками: 300, 500, 700, 900, 1500, 3000 п.н. и 400, 600, 800, 1000, 1400, 2000 и 3000 п.н. соответственно, где верхние полосы размером в 3 т.п.н. в обоих случаях принадлежали векторной плазмиде. Год спустя была опубликована аналогичная работа [Chen et al., 2009] с той лишь разницей, что созданные рекомбинантные плазмиды при расщеплении давали полосы ДНК более крупного размера - 500, 750, 1000, 2000 и 3000 п.н., формируя маркерную лестницу иного предназначения. В своей следующей работе [Wu, Ye, 2011] этими авторами были описаны новые рекомбинатные плазмиды, несущие набор вставок, но общий принцип генерации маркерных лестниц остался прежним.
В литературе также сообщается о создании маркерных лестниц путем клонирования в разных плазмидах по отдельности фрагментов ДНК размером 200, 400, 800 и 1600 п.н. [Planken et al., 2005]. Однако для использования такой маркерной лестницы авторам пришлось осуществлять рестриктазное расщепление всех плазмид, затем проводить препаративный гель-электрофорез с последующей элюцией нужных фрагментов, которые после очистки смешивали в нужной пропорции.
Имеются одна заявка и серия патентов США, выданных разным американским фирмам, в которых описывается создание генно-инженерных конструкций, содержащих в составе одной плазмиды разноразмерные (100, 200, 400, 800, 1200 и 2000 п.н.) фрагменты, которые можно расщепить одной рестриктазой [Hartley, 1998, 2004, 2006, 2010, 2011].
Помимо рестрикционных эндонуклеаз маркерные лестницы ДНК могут изготавливаться с помощью ДНК лигаз. Множественное (контролируемое) лигирование двуцепочечных фрагментов ДНК с образованием смеси фрагментов разных размеров служит удобным способом получения маркеров длин молекул ДНК относительного небольшого размера. Данная технология создания маркерных лестниц описана в патенте США на примере лигирования фрагмента ДНК размером 20 п.н., обеспечивающем формирование ступенек до 1000 п.н. [Singer, 1998]. На основе этого принципа в настоящее время производится немало маркерных лестниц со ступеньками от 5 до 100 п.н.
Abdel-Fattakh с сотр. подобрали 11 праймеров (один прямой и десять - обратные), отжигающихся на разном расстоянии на плазмидном векторе рЕТ-17 и обеспечивающих наработку ампликонов с размерами 100, 198, 304, 399, 500, 750, 1000, 2000 и 2500 п.н. [Abdel-Fattakh, Gaballa, 2006]. Они составили маркерную лестницу ДНК с увеличивающимися кверху ступенями. Индийские авторы [Chandrasekhar et al., 2008] для создания маркерной 100 п.н. лестницы в диапазоне от 100 до 1000 п.н. подобрали один прямой и всего 5 обратных праймеров, которые отжигались на двух плазмидах, одна из которых представляла собой холостой вектор, а другая содержала вставку размером 504 (500) п.н., что позволило при амплификации обеих плазмид образовать на такой лестнице все ступеньки в 100 п.н. без пропусков. В работе авторов из Тайваня [Chang et al., 2008] описан несколько отличающийся подход: одной парой праймеров, подобранных к фланкирующим полилинкер участкам плазмиды pGEM-T Easy, амплифицировали десять различных клонов, несущих вставки от 0 до 900 п.н., обеспечивающих наработку ампликонов с размерами 100, 200, 300, 400, 500, 600, 700, 800, 900 и 1000 п.н. Иранские авторы [Saidijam et al., 2011; Mostaan et al., 2015] также создали серию из 11 рекомбинантных плазмид, содержащих вставки размером от 102 п.н. до 1398 п.н., которые вкупе с холостой плазмидой pTZR57R с помощью ПЦР с одной парой праймеров использовали для наработки «ступенек» лестницы от 100 до 1500 п.н. Для образования полос ДНК размерами 6, 8 и 10 т.п.н. они производили линеаризацию подходящих рекомбинатных плазмид. Вектор pGEM-T Easy, не содержащий вставок для создания маркерной лестницы, использовали таиландские авторы [Riyajan et al., 2011]. С помощью комбинаций из семи прямых и обратных праймеров им удалось образовать ступеньки в 150, 300, 375, 500, 700, 1000, 1200 и 1625 п.н. из последовательности самого вектора.
В 2010 г. другая группа индийских авторов [Gopalakrishnan, Sellappa, 2010] с помощью мультиплексной ПЦР создала разноступенчатую маркерную лестницу, использовав в качестве матрицы ДНК фага лямбда. Они выбрали для мест отжига одного прямого и 6 обратных праймеров участок фага в позициях от 1216 до 2136 п.н., получив в итоге маркерную лестницу со ступеньками, соответствующими 100, 200, 400, 600, 800 и 1000 п.н. Китайскими исследователями [Wang et al., 2010; 2011] создана аналогичная маркерная 100 п.н. лестница, имеющая одинаковые ступеньки от 100 до 1000 п.н. Этими авторами также была выбрана ДНК фага лямбда, только амплифицируемый с помощью десяти прямых и одного обратного праймеров участок располагался в позициях между 6631 и 7630 п.н. Почти тот же участок фага лямбда (7131-7630 п.н.) был использован для амплификации пяти фрагментов 100, 200, 300, 400 и 500 п.н. [Dawson, 1998].
Маркерную лестницу с еще более крупными ступеньками соорудили еще одни китайские авторы [Zhang et al., 2012]. Так, ими были подобраны праймерные пары, ограничивающие при амплификации фрагменты фага лямбда размером 1, 2, 3 и 4 т.п.н. Фрагменты большего размера в их лестнице (5, 6, 8 и 10 т.п.н) представляли собой линеаризованные рекомбинантные плазмиды, несущие вставки соответствующих размеров. Маркерную лестницу с маленькими ступеньками размером всего 6 п. н. разработали испанские авторы [Amills et al., 1996]. Они подобрали 9 праймеров (5 прямых, отжигающихся со сдвигом на 6 п.н., и 4 обратных, также отжигающихся со сдвигом, но уже на 30 п.н.), что в итоге обеспечило создание лестницы с размерами полос от 90 до 204 п.н.
Для проведения различных вариантов пульсирующего гель-электрофореза также существуют маркерные лестницы крупного размера на основе фага лямбда, имеющего длину в 48,5 т.п.н. Обращение с такими протяженными молекулами ДНК требует особой осторожности, и во избежание их порывов такие маркеры еще на стадии приготовления заключают в агарозные блоки, которые потом помещают в лунки геля. Так, описано создание за счет нагрева и охлаждения маркерных лестниц в виде обычных конкатемеров фага лямбда [Waterbury, Lane, 1987] и сшивкой последовательностей этого фага путем дотирования ДНК лигазой фага Т4 с последующей рестрикцией подходящими рестриктазами в условиях неполного расщепления [Cooney, 1992], охватывающими диапазон длин от 48,5 до 1200 т.п.н. и от 48,5 до 200 т.п.н. соответственно. При проведении пульсирующего гель-электрофореза для использования в качестве маркерной лестницы было предложено создавать мультимеры плазмидной ДНК с размерами от 12 до 170 т.п.н. [Hanlon et al., 1989]. Еще одним типом маркерных лестниц следует считать аллельные лестницы, используемые при ДНК-идентификации личности с помощью STR-локусов [Puers et al., 1993; 1994; Schumm, Sprecher, 2006; Green et a., 2013].
В работе [Chang et al., 2008] говорится о принципиальной возможности генерации с помощью транскрипции in vitro под действием подходящих фаговых РНК полимераз маркерных РНК-лестниц с этими же размерами. Было также показано, что с помощью транскрипции с использованием фаговой РНК полимеразы для наработки молекул РНК разной длины для их применения в качестве маркеров длин при гель-электрофорезе можно обойтись и одним вектором со вставкой, если производить его линеаризацию различными рестрикционными эндонуклеазами, сайты которых располагаются на различном удалении от старта транскрипции [Liggit et al., 1994; Audic et al., 1997].
Альтернативой сложному конструированию рекомбинантных плазмид служит транскрипция по типу катящегося кольца по специальной кольцевой ДНК-матрице, которая может иметь разные размеры и быть приготовлена тем или иным способом. Показано, что под действием Т7 РНК полимеразы или РНК полимеразы E. coli могут нарабатываться молекулы РНК длиной свыше 7,5 тысяч нуклеотидов, представляющие собой конкатемерные повторы, мономерная единица которых равна по размеру исходному кольцу [Daubendiek, Kool, 1997; Diegelman et al., 1998]. Наличие в этих кольцах участков, кодирующих cis-рибозимы, приводит к расщеплению первичного транскрипта на фрагменты РНК, формирующие на картине гель-электрофореза характерную лестницу полос с одинаковыми ступенями. В работе китайских авторов [Wang et al., 2014] помимо рибозимного расщепления РНК-конкатемеры, образующиеся в результате транскрипции катящимся кольцом, предложено также фрагментировать с помощью РНКазы Н и модифицированного фрагмента ДНК, отжигающегося в нужных местах первичного транскрипта.
В настоящее время в каталогах множества фирм, специализирующихся на поставке реактивов для молекулярной биологии, представлены многочисленные маркерные лестницы, несущие фрагменты ДНК или РНК широкоразмерного диапазона, изготовленные разными способами. Среди них несколькими фирмами поставляется ставшая классикой маркерная лестница 123 bp Ladder. Очень большую линейку таких маркеров поставляет группа фирм, входящих в концерн Thermo Fisher Scientific. Именно этим фирмам принадлежит большая часть патентов, в которых описаны способы изготовления различных маркерных ДНК-лестниц. Например, маркерные лестницы GeneRuler, O'GeneRuler, MassRuler, FastRuler, ZipRuler Express представляют собой смеси различных фрагментов ДНК, каждый из которых очищен хроматографически. За счет индивидуальной очистки каждого фрагмента существует возможность изготовления специалистами этой фирмы маркерных лестниц NoLimits с любыми (из существующего перечня) ступеньками «под заказ» для заинтересованных в этом пользователей, решающих конкретные узконаправленные задачи. Маркерные лестницы O'RangeRuler содержат наборы молекул ДНК, представляющих собой результат неполного (частичного) лигирования тупоконечных фрагментов ДНК с повторяющимися единицами с размерами от 5 до 500 п.н. Помимо ДНК-лестниц, этой фирмой производятся также РНК-лестницы RiboRuler, представляющие собой смесь из хроматографически очищенных транскриптов в виде 8 ступенек для High Range (от 100 до 6000 нуклеотидов) и 7 ступенек для Low Range (от 100 до 1000 нуклеотидов).
Фирма New England Biolabs, Inc. поставляет почти два десятка различных маркерных лестниц для гель-электрофореза как ДНК, так и РНК. Причем диапазон длин транскриптов в РНК-лестницах довольно велик, от 17 нуклеотидов (для детекции микроРНК) до 9000 нуклеотидов. Отдельно стоит упомянуть лестницу Reverse Mass DNA Ladder, в которой фрагменты меньшего размера представлены в большем количестве, чем все остальные, тогда как для многих маркерных лестниц нижние ступеньки бывают видны в геле крайне плохо. Кроме линейных маркерных молекул ДНК фирма New England Biolabs, Inc. поставляет лестницу из 9 суперскрученных плазмид с размерами от 2 до 10 т.п.н. Для пульсирующего гель-электрофореза среди прочих маркеров продается лестница, представляющая собой конкатемеры фага лямбда, покрывающие диапазон от 50 т.п.н. до 1 млн.п.н. Также этой фирме принадлежит товарный знак TriDye DNA Ladder, под которым скрывается продукт, представляющий смесь трех лидирующих красителей: ксиленцианола, бромфенолового синего и оранжевого G, мигрирующих в стандартном 1%-ном агарозном геле рядом с фрагментами ДНК длиной 4 т.п.н., 300 п.н. и 50 п.н. соответственно. TriDye DNA Ladder предназначен не для точного установления размеров разделяемых фрагментов, а служит лишь ориентиром продолжительности гель-электрофореза, хотя на заре злектрофоретического анализа ДНК эти красители рекомендовалось использовать для определения длин ДНК.
Таким образом, на сегодняшний день описано множество способов получения маркерных лестниц, а на рынке представлен широкий набор коммерческих продуктов. Однако большую часть коммерчески доступных маркерных лестниц получают преимущественно путем рестриктазного расщепления фагов и плазмид. Используемый способ производства обусловливает относительно высокую стоимость таких продуктов. Кроме того, размеры "ступенек" в подобных маркерных лестницах зависят от используемых рестриктаз и расположения их сайтов узнавания.
Суть предлагаемого способа заключается в следующем. Маркерные лестницы получаются с помощью реакции амплификации "катящимся кольцом" (АКК, англ. RCA -rolling circle amplification) в варианте разветвленной АКК, или рамификации (англ. ramification). В качестве матрицы используется кольцевая молекула одноцепочечной ДНК требуемого размера L (где L - количество нуклеотидов), которая образуется из синтетического олигонуклеотида размера L путем внутримолекулярного лигирования с помощью Т4 РНК лигазы (фиг. 1, реакция 1) или любой другой лигазы, способной к внутримолекулярному лигированию одноцепочечных нуклеиновых кислот. В ходе реакции образуются преимущественно линейные продукты лигирования, представляющие собой конкатемерные (повторяющиеся) структуры, которые в дальнейшем не мешают получению маркерных лестниц. Необходимым условием высокого выхода целевого продукта лигирования (кольца) является наличие в реакционной смеси достаточного количества (5-7%) полиэтиленгликоля (ПЭГ); наиболее удобен в применении ПЭГ со средней молекулярной массой 4000-6000 Да, но можно использовать также ПЭГ с другими молекулярными массами и другие инертные загустители, например, многоатомные спирты (глицерин и т.п.), олигосахара и пр. (пример 1).
На следующем этапе кольцевой продукт лигирования используется в качестве матрицы для отжига и элонгации праймера 1, выступающего в качестве "затравочного" праймера (фиг. 1, реакция 2), а присутствующий в реакционной смеси второй праймер (праймер 2) обеспечивает запуск АКК по типу рамификации (фиг. 1, реакция 3). Наличие двух праймеров обеспечивает наработку двуцепочечных фрагментов ДНК, кратных по размеру кольцевой матрице, (т.е. nL, где n - число оборотов полимеразы по кольцу). Визуализация результатов рамификации методом гель-электрофореза дает характерную лестницу полос (фиг. 2).
Для реакции полимеризации полинуклеотидной цепи, в зависимости от вида получаемой маркерной лестницы (ДНК или РНК), можно использовать ДНК или РНК полимеразы, обладающие цепь-смещающей активностью, например, ДНК-полимеразы группы Bst (большой фрагмент Bst, или Bst exo-, Bst 2.0, Bst 2.0 WarmStart, Bst 3.0 (New England Biolabs)), Vent exo-, Deep Vent, 9°Nm, Bsm exo-, Aac и т.п. и T7 РНК полимеразу. В случае термолабильных ДНК-полимераз (Bst exo-, Bst 2.0, Bst 2.0 WarmStart, Bst 3.0, Bsm exo-, Aac) амплификацию проводят в изотермическом режиме при температуре, близкой к температурам максимума активности фермента и отжига праймеров (пример 2). Внесение фермента осуществляют после проведения предварительной денатурации и отжига праймера, хотя данный этап не является обязательным и служит только для повышения выхода и качества продуктов амплификации. Термостабильные ДНК полимеразы (Vent exo-, Deep Vent, 9°Nm) вносятся сразу в реакционную смесь, далее следует конечное число циклов изменения температуры, после чего выдерживают смесь при постоянной температуре (пример 3). Термоциклирование необходимо для создания протяженных мишеней путем денатурации исходных и образующихся матриц и отжига праймеров, а выдерживание при постоянной температуре - для повышения выхода продуктов реакции. При этом возможно использование для амплификации одного только олигонуклеотида - предшественника кольцевой матрицы (пример 4), который также будет обеспечивать наработку необходимых фрагментов, но с меньшей эффективностью.
При добавлении в реакционную смесь небольшого количества рестриктаз или никаз можно добиться расщепления растущих цепей нуклеиновых кислот по сайтам узнавания данных ферментов, предварительно заданных в нуклеотидной последовательности кольцевой матрицы, тем самым снизив количество очень протяженных продуктов амплификации в пользу более коротких (пример 5). При правильном соотношении полимеразы и эндонуклеазы достигается желаемый профиль продуктов и, соответственно, картина полос в маркерной лестнице (фиг. 3).
Для получения РНК-маркерных лестниц применяется ДНК-кольцевая матрица, рибо- и дезоксирибонуклеозидтрифосфаты и две обладающие цепь-смещающей активностью полимеразы: РНК полимераза фага Т7 и ДНК полимераза Bst 3.0 (пример 6). Необходимым условием протекания амплификации в этом случае является наличие в праймере 1 по 5'-концу нуклеотидной последовательности Т7 промотора -TAATACGACTCACTATAG. Поскольку Т7 РНК полимераза строит РНК-цепь по цепи ДНК, при амплификации катящимся кольцом она будет нарабатывать только одноцепочечный РНК-продукт, а рамификация (образование набора конкатемерных продуктов) протекать не будет несмотря на наличие второго праймера. В связи с этим необходимо внесение дополнительно ДНК полимеразы Bst 3.0, которая обладает уникальной способностью строить цепи ДНК как по ДНК, так и по РНК матрице. В указанном варианте проведения реакции происходит одновременное образование и ДНК, и РНК-цепей, что позволяет безостановочно работать обоим ферментам благодаря наличию постоянно синтезирующихся подходящих матриц (фиг. 4). Итогом реакции является набор фрагментов, представляющих собой два вида двуцепочечных структур: ДНК-ДНК (дцДНК) и ДНК-РНК, а двуцепочечные РНК-РНК не образуются. При правильном количественном соотношении ферментов и дезоксирибо- и рибонуклеозидтрифосфатов ДНК-РНК гибриды составляют основную долю молекул. Для получения конечной РНК-маркерной лестницы препарат сначала очищают от полимераз, праймеров и трифосфатов, а затем подвергают обработке ДНКазой I, способной расщеплять дцДНК и ДНК-РНК гибриды по ДНК-части, и последующей очистке. В итоге получают набор одноцепочечных РНК-фрагментов, размеры которых не будут кратны размеру исходной матрицы за счет наличия 5'-«хвостовой» последовательности, что необходимо учитывать при конструировании кольцевой матрицы. Возможен вариант, когда нуклеотидная последовательность сайта узнавания Т7 РНК полимеразы задается в структуре кольцевой матрицы. В этом случае праймер 1 не будет иметь некомплементарной 5'-концевой части, и размеры РНК-фрагментов будут кратны размеру исходной матрицы.
Для получения маркерных лестниц с размерами фрагментов, не кратными размеру кольцевой матрицы, можно использовать праймеры с 5'-концевой («хвостовой») последовательностью любой требуемой длины и некомплементарной исходной кольцевой матрице (фиг. 5). 5'-«Хвостовую» последовательность может содержать только один из праймеров или оба. Варьируя ее длину, можно создавать маркерные лестницы с любым размером шага с точностью до единичного нуклеотида, учитывая, что при амплификации к длине кольца прибавляются размеры 5'-«хвостовой» последовательности (пример 7). При этом размеры фрагментов определяются уравнением n⋅(L+M+N), где L - размер кольцевой матрицы, М - длина 5'-концевой последовательности первого праймера, N - длина 5'-концевой последовательности второго праймера, a n - число оборотов полимеразы вокруг кольца.
Другим способом получения маркерных лестниц с размерами фрагментов, не кратными размеру кольцевой матрицы, является простое смешивание препаратов, полученных при амплификации кольцевых матриц разного размера (пример 8). Например, если взять две маркерные лестницы с разными длинами фрагментов: одна 50, 100, 150, 200, 250, 300, 350 и т.д., вторая 75, 150, 225, 300, 375 и т.д., то получится маркерная лестница со следующими длинами фрагментов: 50, 75, 100, 150, 200, 225, 250, 300, 350, 375 и т.д.
Получаемые после АКК реакционные смеси можно подвергать очистке методами фенольно-хлороформной экстракции по известным протоколам и/или с помощью коммерческих наборов, предназначенных для выделения ДНК и РНК из реакционных смесей. Конечные препараты маркерных лестниц должны храниться при -20°С, недопустимо их выдерживание при температуре выше +40°С. Использование маркерных лестниц, полученных описанным выше способом, для гель-электрофоретического анализа нуклеиновых кислот идентично таковому для всех коммерчески доступных маркерных лестниц (пример 9).
Изобретение иллюстрируется следующими конкретными примерами.
Пример 1. Получение одноцепочечной кольцевой матрицы.
Одноцепочечную кольцевую матрицу получают из линейного синтетического олигодезоксирибонуклеотида (ОДН) любой требуемой длины L. ОДН подвергают лигированию с помощью Т4 РНК лигазы, например, по следующей методике. К 1 мкл раствора, содержащего 0.1 О.Е. ОДН, добавляют 2 мкл буфера для лигазы, 3.0 мкл ПЭГ6000, 2 мкл 10 мМ АТФ, 5 ед. акт. Т4 РНК лигазы или любой другой лигазы, способной к внутримолекулярном) лигированию одноцепочечных нуклеиновых кислот, и доводят объем реакционной смеси до 20 мкл, инкубируют 18 ч при температуре 8-15°С и затем прогревают 10 мин при 75°С. Полученную кольцевую матрицу можно далее использовать для реакций амплификации без дополнительной очистки или подвергать очистке любыми известными способами.
Пример 2. Получение ДНК-маркерных лестниц в режиме изотермической амплификации.
Готовят 20-50 мкл реакционной смеси для амплификации, содержащей 1 мкл раствора одноцепочечной кольцевой матрицы, по 1-2 мкл каждого из праймеров 1 и 2 (концентрация 2.0 О.Е./мл), 2-5 мкл смеси дезоксинуклеозидтрифосфатов (dNTP) с концентрацией 2.5 мМ, 2-5 мкл соответствующего буфера для ДНК-полимеразы. Используют следующий протокол амплификации. Сначала прогревают образцы 3 мин при 95°С (денатурация), затем плавно снижают температуру до расчетной температуры отжига праймеров (оптимально 50-60°С), после чего вносят 1-5 ед. акт. одной из ДНК-полимераз группы Bst (Bst exo-, Bst 2.0, Bst 3.0) или любой другой термолабильной ДНК-полимеразы с цепь-смещающей активностью и выдерживают реакционную смесь 2 ч при температуре отжига праймеров. Далее проводят выделение продуктов амплификации широко используемым методом фенольно-хлороформной экстракции.
Пример 3. Получение ДНК-маркерных лестниц в режиме квазиизотермической амплификации.
Готовят 20-50 мкл реакционной смеси для амплификации, содержащей 1 мкл раствора одноцепочечной кольцевой матрицы, по 1-2 мкл каждого из праймеров 1 и 2 (концентрация 2.0 О.Е./мл), 2-5 мкл смеси dNTP с концентрацией 2.5 мМ, 1-5 ед. акт. ДНК-полимеразы Vent exo- или любой другой термостабильной ДНК-полимеразы с цепь-смещающей активностью, 2-5 мкл соответствующего буфера для ДНК-полимеразы. Используют следующий протокол амплификации. Сначала прогревают образцы 3 мин при 95°С (денатурация), затем проводят 15-20 циклов термоциклирования (95°С - 30 с, 50-60°С (в зависимости от расчетной температуры отжига праймеров) - 30 с, 75°С - 30 с), и затем выдерживают реакционную смесь 2 ч при температуре отжига праймеров. Далее проводят выделение продуктов амплификации аналогично примеру 2.
Пример 4. Получение ДНК-маркерных лестниц без использования праймеров.
Готовят реакционные смеси для амплификации согласно примерам 2 или 3, но отличающиеся отсутствием праймеров и наличием синтетического олигонуклеотида - предшественника кольцевой матрицы. Используют протоколы амплификации и очистки, описанные в примерах 2 и 3.
Пример 5. Получение ДНК-маркерных лестниц, совмещенное с эндонуклеазным расщеплением.
Готовят реакционные смеси для амплификации согласно примерам 2 или 3, но отличающиеся наличием в них дополнительно соответствующего буфера в необходимой концентрации для любой подходящей рестриктазы или любой подходящей никазы, обеспечивающих расщепление цепей ДНК по сайтам узнавания, предварительно заданных в нуклеотидной последовательности кольцевой матрицы. Используют протоколы амплификации, описанные в примере 2 или 3, но отличающиеся тем, что внесение 1-5 ед. акт. рестриктазы или никазы осуществляют одновременно с ДНК-полимеразой. Выделение продуктов проводят аналогично примеру 2.
Пример 6. Получение РНК-маркерных лестниц.
Готовят 20-50 мкл реакционной смеси для амплификации, содержащей 1 мкл раствора одноцепочечной кольцевой матрицы, по 1-2 мкл каждого из праймеров (концентрация 2.0 О.Е./мл), несущих промотор Т7 РНК полимеразы, 3 мкл смеси рибонуклеозидтрифосфатов (NTP) с концентрацией 2.5 мМ, 1 мкл смеси дезоксирибонуклеозидтрифосфатов (dNTP) с концентрацией 2.5 мМ, 3 мкл буфера для Т7 РНК-полимеразы и 3 мкл буфера для ДНК полимеразы Bst 3.0. Используют следующий протокол амплификации. Сначала прогревают образцы 3 мин при 95°С (денатурация), затем плавно снижают температуру до 40-45°С, после чего вносят 10 ед. акт. Т7 РНК-полимеразы, 1 ед. акт. ДНК полимеразы Bst 3.0 и выдерживают реакционную смесь 3,5-4,0 ч при 40-45°С. Далее проводят очистку продуктов амплификации с использованием любых реагентов для выделения РНК, например, Trizol. К полученному препарату добавляют 2 ед. акт. ДНКазы I и 5 мкл соответствующего буфера из расчета на 50 мкл реакционной смеси, выдерживают 30 мин при 37°С, далее вновь проводят очистку с использованием любых реагентов для выделения РНК.
Пример 7. Получение маркерных лестниц с шагом, отличным от размера кольцевой матрицы.
Получение маркерных лестниц с шагом, отличным от размера кольцевой матрицы, осуществляют аналогично методикам, описанным в примерах 2-6, отличающихся наличием в реакционной смеси праймеров с 5'-концевой последовательностью любой требуемой длины и некомплементарной исходной кольцевой матрице.
Пример 8. Получение маркерных лестниц со смешанными длинами фрагментов.
Получение маркерных лестниц со смешанными длинами фрагментов осуществляют с помощью простого смешения препаратов, полученных при амплификации кольцевых матриц разного размера согласно примерам 2-5.
Пример 9. Проведение гель-электрофоретического анализа нуклеиновых кислот с помощью маркерных лестниц.
Электрофорез ДНК (например, при анализе результатов амплификации, рестрикции, лигирования и т.п.), а также РНК (например, после выделения РНК из биологических образцов), проводят с использованием полиакриламидных, агарозных или смешанных гелей в любом стандартном буфере в камерах вертикального или горизонтального типа. Гели окрашивают любым из известных методов обнаружения нуклеиновых кислот в гелях и визуализируют в соответствующих приборах.
Литература, принятая во внимание
1. Abdel-Fattah Y.R., Gaballa A. Synthesis of DNA ladder by polymerase chain reaction and optimization of yield using surface // Biotechnology. 2006. V. 5. P. 166-172.
2. Amills M., Francino O., Sánchez A. Primer-directed synthesis of a molecular weight marker // Genet. Anal. 1996. V. 13. P. 147-149.
3. Barvish Z., Davis C, Gitelman I. A wide-range, low-cost 150 bp ladder for sizing DNA fragments between 150 and 4500 bp // Electrophoresis. 2007. V. 28. P. 900-902.
4. Chandrasekhar N., Vijayan N.N., Vaidyan L.K., Mathew A., Srinivas L., Banerjee M. Universal protocol for generating 100 bp size standard for endless usage // Electronic Journal of Biotechnology 2008. V. 11. No. 2, DOI: 10.2225/vol11-issue2-fulltext-10.
5. Chang M., Wang J.H., Lee H. J. Laboratory production of 100 base pair DNA molecular weight markers // J. Biochem. Biophys. Methods. 2008. V. 70. P. 1199-1202.
6. Chen Z., Wu J., Li X., Ye C, Wenxing H. Novel strategies to construct complex synthetic vectors to produce DNA molecular weight standards // Mol. Biotechnol. 2009. V. 42. P. 128-133.
7. Cooney C.A. Separation and size determination of DNA over a 10-200 kbp Range // Methods Mol. Biol. 1992. V. 12. P. 31-37.
8. Dawson E.P. Method for the multiplexed preparation of nucleic acid molecular weight markers and resultant products / US Patent 5,714,326. Filed: Dec. 5, 1994. Issued: Feb. 3, 1998.
9. Dongyi H., Longhai Z., Huazong Z., Ye C. Construction of DNA marker plasmids based on Taq tailing activity and selective recovery of ligation products // Plant Mol. Biol. Reporter. 2008. V. 26. P. 316-323.
10. Gopalakrishnan R., Joseph S., Sellappa S. Constructing a DNA ladder Range for Lambda Phage by multiplex PCR // Iran J. Microbiol. 2010. V. 2. P. 210-212.
11. Green R., Mulero J., Hennessy L., Lagace R., Chang C.W. Allelic ladder loci / US Patent 8,409,806. Filed: Nov. 24, 2010. Issued: Apr. 2, 2013.
12. Hanlon D.J., Smardon A.M., Lane M.J. Plasmid multimers as high resolution molecular weight standards for pulsed field gel electrophoresis // Nucleic Acids Res. 1989. V. 17. P. 5413.
13. Hartley J.L. Nucleic acid marker ladder for estimating mass / US Patent 5,834,201. Filed: Jul. 11, 1997. Issued: Nov. 10, 1998.
14. Hartley J.L. Nucleic acid marker ladder for estimating mass / US Patent 6,680,378. Filed: Sep.20, 2000. Issued: Jan. 20, 2004.
15. Hartley J.L. Nucleic acid marker ladder for estimating mass. US Patent 7,132,520. Filed: Apr. 10, 2003. Issued: Nov. 7, 2006.
16. Hartley J.L. Nucleic acid marker ladder for estimating mass / US Patent 7,846,665. Filed: Sep.25, 2008. Issued: Dec. 7, 2010.
17. Hartley J.L. Nucleic acid marker ladder for estimating mass / U.S. Patent Application Publication 2011/0132760 A1. Filed: Dec. 7, 2011. Pub. Date: Jun. 9, 2011.
18. Hartley J.L., Gregori T.J. Cloning multiple copies of a DNA segment // Gene. 1981. V. 13. P. 347-353.
19. Hartley J.L., Gregori T.J. Genetic reagents for generating plasmids containing multiple copies of DNA segments / US Patent 4,403,036. Filed: Dec. 2, 1980. Issued: Sep.6, 1983.
20. Hu A.W., Hartley J.L., Jordan H.J. Nucleic acid ladders / US Patent 6,924,098. Filed: Dec. 23, 1999. Issued: Aug. 2, 2005.
21. Hyman E.D. DNA ladders / US Patent Number 5,840,575. Filed: Jan. 10. 1997. Issued: Nov. 24, 1998.
22. Lan V.T., Loan P.T., Duong P.A., Thanh E.T., Ha N.T., Thuan T.B. Straightforward procedure for laboratory production of DNA ladder // J. Nucleic Acids. 2012. V. 2012. ID 254630.
23. Maniatis Т., Jeffrey A., van deSande H. Chain length determination of small double- and single-stranded DNA molecules by polyacrylamide gel electrophoresis // Biochemistry. 1975. V. 14. P. 3787-3794.
24. Minter S., Sealey P.G. Nucleic acid molecular weight markers. Appendix 1. In: Gel Electrophoresis on Nucleic Acids. A Practical Approach. (D. Rickwood, B.D. Hames, eds.) IRL Press. Oxford, Washington DC. 1982. P. 227-232.
25. Mostaan S., Ajorloo M., Khanahmad H., Cohan R.A., Tehrani Z.R., Rezaei M., Fazeli F., Behdani M., Zare S.K., Karimi Z., Mozhgani S.H., Moukhah R. A novel combined method for cost-benefit production of DNA ladders // Adv. Biomed. Res. 2015. V. 4. P. 15.
26. Murray K., Murray N.E. Phage lambda receptor chromosomes for DNA fragments made with restriction endonuclease III of Haemophilus influenzae and restriction endonuclease I of Escherichia coli // J. Mol. Biol. 1975. V. 98. P. 551-564.
27. Planken K.L., Koenderink G.H., Roozendaal R., Philipse A.P. Monodisperse DNA restriction fragments I. Synthesis and characterization // J. Coll. Interface Sci. 2005. V. 291. P. 120-125.
28. Puers C., Hammond H.A., Jin L., Caskey C.T., Schumm J.W. Identification of repeat sequence heterogeneity at the polymorphic short tandem repeat locus HUMTH01 [AATG]n and reassignment of alleles in population analysis by using a locus-specific allelic ladder // Am. J. Hum. Genet. 1993. V. 53. P. 953-958.
29. Puers C., Hammond H.A., Caskey C.T, Lins A.M., Sprecher C.J., Brinkmann В., Schumm J.W. Allelic ladder characterization of the short tandem repeat polymorphism located in the 5'-flanking region to the human coagulation factor XIII A subunit gene // Genomics. 1994. V. 23. No. 1. P. 260-264.
30. Riyajan S., Yentua W., Phunchongsakuldit J. Low cost DNA molecular weight magrker: primer-directed synthesis from pGEM-T easy vector // Walailak J. Sci. Tech. 2011. V. 8. P. 187-192.
31. Saidijam M., Shanreza H.K., Tehrani Z.R., Karimizare S., Shabab N., Behdani M. Designing and constructing an 100 bp DNA ladder by combining PCR and enzyme digestion methods // Tehran Univ. Med. J. 2011. V. 69. P. 75-82.
32. Sanger F., Coulson A.R., Hong G.F., Hill D.F., Petersen G.B. Nucleotide sequence of bacteriophage lambda DNA // J. Mol. Biol. 1982. V. 162. P. 729-773.
33. Schumm J.W., Sprecher C.J. Multiplex amplification of short tandem repeat loci / Patent Number US 7,008,771. Filed: Sep.6, 2002. Issued: Mar. 7, 2006.
34. Sealey P.G., Southern E.M. Gel electrophoresis of DNA / In: Gel Electrophoresis on Nucleic Acids. A Practical Approach. (D. Rickwood, B.D. Hames, eds.) IRL Press. Oxford, Washington DC. 1982. P. 39-76.
35. Singer P.A. Polynucleotide sizing reagents / US Patent Number 5,824,787. Filed: Feb. 2, 1996. Issued: Oct. 20, 1998.
36. Southern E. Gel electrophoresis of restriction fragments. Methods Enzymol. 1979. V. 68. P. 152-176.
37. Sutcliffe J.G. pBR322 restriction map derived from the DNA sequence: accurate DNA size markers up to 4361 nucleotide pairs long // Nucleic Acids Res. 1978. V. 5. P. 2721-2728.
38. Sutcliffe J.G. Complete nucleotide sequence of the Escherichia coli plasmid pBR322 // Cold Spring Harb. Symp.Quant. Biol. 1979. V. 43. P. 77-90.
39. Wang T.Y., Guo L., Zhang J.H. Preparation of DNA ladder based on multiplex PCR technique // J. Nucleic Acids. 2010. V. 2010. ID 421803.
40. Wang T.Y., Wang L., Wang F. Methodology: simplified preparation of a DNA ladder using PCR // Genet. Mol. Res. 2011. V. 10. P. 1631-1635.
41. Waterbury P.G., Lane M.J. Generation of lambda phage concatemers for use as pulsed field electrophoresis size markers // Nucleic Acids Res. 1987. V. 15. P. 3930.
42. Wu J., Ye C. Tandem PCR: a novel and efficient unit amplification model for the preparation of small DNA fragments // Mol. Biol. Rep.2011. V. 38. P. 2729-2731.
43. Ye C., Gu J., Chen S., Deng A., Li Y.Z., Li D. Unit cloning and amplification as novel and universal strategies for complex vector construction and small DNA fragment preparation // Electrophoresis. 2010. V. 31. P. 2929-2935.
44. Zhang J.H., Yang R., Wang T.Y., Dong W.H., Wang F., Wang L. A simple and practical method that prepares high molecular weight DNA ladders // Mol. Med. Rep.2012. V. 6. P. 1211-1213.

Изобретение относится к биохимии. Описан способ получения специального вида расходных материалов для медико-биологических исследований - маркеров длин фрагментов нуклеиновых кислот (ДНК и РНК маркерных лестниц). Сущность изобретения заключается в наработке фрагментов ДНК и РНК, составляющих лестницу, с помощью реакции амплификации "катящимся кольцом" в варианте рамификации. В качестве матрицы для амплификации используется кольцевая молекула одноцепочечной ДНК требуемого размера L (где L - количество нуклеотидов), которая образуется из синтетического олигонуклеотида размера L путем внутримолекулярного лигирования. Кольцевой продукт лигирования используется в качестве матрицы для отжига и элонгации "затравочного" праймера 1, а присутствующий в реакционной смеси праймер 2 обеспечивает запуск амплификации по типу рамификации. Для реакции полимеризации полинуклеотидной цепи используются ДНК или РНК полимеразы, обладающие цепь-смещающей активностью, амплификацию проводят в изотермическом или квазиизотермическом режиме. При добавлении в реакционную смесь небольшого количества рестриктаз или никаз можно добиться расщепления растущих цепей нуклеиновых кислот по сайтам узнавания данных ферментов, тем самым снизив количество очень протяженных продуктов амплификации в пользу более коротких. Для получения РНК-маркерных лестниц применяются дезоксирибо- и рибонуклеозидтрифосфаты и смесь ДНК и РНК полимераз. Для получения маркерных лестниц с размерами фрагментов, не кратными размеру кольцевой матрицы, используются праймеры с 5'-концевой последовательностью любой требуемой длины и некомплементарной исходной кольцевой матрице. Другим способом получения маркерных лестниц с размерами фрагментов, не кратными размеру кольцевой матрицы, является простое смешивание препаратов, полученных при амплификации кольцевых матриц разного размера. Предлагаемый способ позволяет получать маркерные лестницы в значительном количестве с любым размерным шагом. Они могут быть использованы медицинскими диагностическими лабораториями, лабораториями экспертизы и научно-исследовательскими лабораториями при проведении всех видов гель-электрофоретического анализа нуклеиновых кислот. Использование маркерных лестниц, полученных описанным выше способом, идентично таковому для всех коммерчески доступных маркерных лестниц. 2 н. и 3 з.п. ф-лы, 5 ил., 9 пр.
1. Способ получения маркеров длин фрагментов ДНК (ДНК-маркерных лестниц), отличающийся тем, что для получения используется реакция амплификации "катящимся кольцом" в варианте рамификации с участием синтетической кольцевой ДНК-матрицы заданного размера, двух праймеров и ДНК полимераз с цепь-смещающей активностью, обеспечивающих образование набора конкатемерных ДНК продуктов определенной длины каждый, кратной размеру исходной кольцевой ДНК-матрицы.
2. Способ получения маркеров длин РНК (РНК-маркерных лестниц), отличающийся тем, что для получения используется реакция амплификации "катящимся кольцом" в варианте рамификации с участием синтетической кольцевой ДНК-матрицы заданного размера, двух праймеров, один из которых содержит сайт узнавания Т7 РНК полимеразы, и смеси из ДНК полимеразы Bst 3.0 и Т7 РНК полимеразы с последующим расщеплением ДНК-цепей с помощью ДНКазы I.
3. Способ по п. 1, отличающийся тем, что для получения маркеров длин фрагментов ДНК в структуре кольцевой матрицы задается сайт узнавания какой-либо рестриктазы или никазы, а в реакционную смесь добавляется соответствующая рестриктаза или никаза, что в результате обеспечивает смещение профиля образующихся продуктов амплификации в сторону более коротких фрагментов.
4. Способ по п. 1, отличающийся тем, что для получения маркеров длин фрагментов ДНК используются праймеры с 5'-концевой последовательностью любой требуемой длины и некомплементарной исходной кольцевой матрице, обеспечивающие образование маркерных лестниц с любым размером шага с точностью до единичного нуклеотида.
5. Способ по п. 1, отличающийся тем, что для получения маркеров длин фрагментов ДНК используются препараты, полученные простым смешиванием реакционных смесей, полученных при амплификации кольцевых матриц разного размера.
| Застежка для обуви и т.п. | 1930 |
|
SU21069A1 |
| US 20010039039 А1, 08.11.2001. | |||
Авторы
Даты
2017-04-13—Публикация
2015-12-28—Подача