Область техники, к которой относится изобретение
Изобретение относится к молекулярной биологии, микробиологии и медицине и обеспечивает ДНК-чип для идентификации генетических детерминант антибиотикорезистентности возбудителей инфекций, приводящих к нарушению репродуктивных функций человека (ИПНРФ): Treponema pallidum, Neisseria gonorrhoeae, Chlamydia trachomatis, Mycoplasma genitalium, Mycoplasma hominis, Gardnerella vaginalis, Ureaplasma parvum, Ureaplasma urealyticum, Atopobium vaginae, Enterococcus faecalis, Trichomonas vaginalis, Escherichia coli, Mobiluncus mulieris, Streptococcus anginosus, Fusobacterium nucleatum, Staphylococcus epidermidis, Bacteroides fragilis, к антимикробным препаратам, включающим антибиотики пенициллинового ряда, цефалоспорины, тетрациклины, фторхинолоны, макролиды, нитроимидазолы, спектиномицин, а также набор олигонуклеотидов для иммобилизации на ДНК-чипе.
Уровень техники
Согласно Международной классификации болезней (МКБ-10, http://www.medicusamicus.com/index.php?action=mkb), к инфекциям, вызывающим заболевания урогенитального тракта и приводящих к нарушениям репродуктивных функций человека, относятся сифилис, гонококковая инфекция, урогенитальный трихомониаз, хламидийная инфекция и пр. Патологические состояния мочеполовой системы часто носят полимикробный характер, представленный не только облигатными патогенами (Neisseria gonorrhoeae, Chlamydia trachomatis, Treponema pallidum, Mycoplasma genitalium), но и условно-патогенными инфекционными агентами (Mobiluncus, Bacteroides, Fusobacterium, Ureaplasma, Streptococci, Staphylococci, Enterococci, E. coli, Gardnerella vaginalis и др.). Серьезной проблемой во всем мире является рост лекарственной устойчивости возбудителей заболеваний, что вызывает значительные затруднения при выборе методов лечения пациентов. В докладе ВОЗ «Устойчивость к противомикробным препаратам: глобальный доклад по эпиднадзору» (апрель 2014 г.) отмечается большая опасность роста устойчивости к антибактериальным препаратам для целого ряда возбудителей инфекций, и среди вызывающих наибольшую озабоченность инфекций, наряду с туберкулезом, малярией, стафилококковой инфекцией, выделены N. gonorrhoeae (появление штаммов с уменьшенной чувствительностью к цефалоспоринам третьего поколения) и Е. coli (устойчивость к фторхинолонам и цефалоспоринам). Таким образом, для выяснения этиологии инфекционно-воспалительного процесса органов мочеполовой системы и последующей успешной терапии необходимо не только выявление в клинических образцах инфекционных агентов, включая одновременную идентификацию большого количества возбудителей, но также анализ чувствительности/устойчивости инфекционных агентов к основным противомикробным препаратам. В силу чрезвычайного разнообразия механизмов устойчивости возбудителей ИПНРФ разрабатываемые методики должны охватывать максимально возможный спектр молекулярных параметров, ассоциированных с резистентностью (Shaskolskiy В, Dementieva Е, Leinsoo A, Runina A, Vorobyev D, Plakhova X, Kubanov A, Deryabin D, Gryadunov D. 2016. Drug resistance mechanisms in bacteria causing sexually transmitted diseases and associated with vaginosis. Frontiers in Microbology. 7:747).
Основными методами лабораторной диагностики заболеваний являются микробиологические (микроскопическое и культуральное исследование) и молекулярно-биологические (полимеразная цепная реакция (ПЦР), NASBA (Nucleic Acids Sequence-Based Amplification) методы исследования, определение нуклеотидной последовательности участков микроорганизмов, в частности, выявление мутаций, ведущих к резистентности к лекарственным препаратам. Микроскопическое исследование является наиболее доступным методом, однако его чувствительность при идентификации ряда инфекционных агентов не превышает 40-60% в связи с субъективной оценкой результатов анализа. Культуральное исследование обладает высокой специфичностью и чувствительностью (96-100%), однако является дорогостоящим и трудоемким, что ограничивает его широкое применение. Культивирование ряда микроорганизмов (М genitalium, Mobiluncus, Peptostreptococcus, Prevotella, Bacteroides, Fusobacterium и др.) невозможно в условиях лабораторий медицинских учреждений в связи с высокой требовательностью к питательным средам и длительностью их культивирования (до 5 месяцев), а Т. pallidum in vitro не культивируется.
Идентификацию микроорганизмов выполняют также с использованием метода масс-спектрометрии, который позволяет проводить анализ не только чистых культур, но и биологического материала. К недостаткам микробиологической масс-спектрометрии относится несовершенство идентификации микроорганизмов в смешанных культурах и отсутствие стандартных критериев в оценке антибиотикорезистентности (Seng Р., Drancourt M. Gouriet F., La Scola В., Fournier P.E., Rolain J.M., Raoult D. Ongoing revolution in bacteriology: routine identification of bacteria by matrix-assisted laser desorption ionization time-of-flight mass spectrometry, Clin Infect Dis, 2009 v. 49, p. 543-551.). Высокая стоимость оборудования и квалификация персонала позволяют использовать данный подход только в крупных централизованных бактериологических лабораториях.
Для определения фенотипической резистентности микроорганизмов к антимикробным препаратам используют методы серийного разведения на агаре и диско-диффузионный метод. «Золотым стандартом» микробиологической диагностики антибиотикорезистентности является метод серийных разведений. Все большую распространенность приобретают автоматизированные системы культивирования микроорганизмов и определения их лекарственной чувствительности, такие как VITEK ( , Франция), ВАСТЕС (Becton Dickinson, США), Sensititre TREK Diagnostic Systems (Thermo Scientific, США). С использованием данных систем можно получить количественную информацию об уровне устойчивости микроорганизмов к различным препаратам, однако время проведения анализа для получения данных о профиле резистентности составляет от 7 суток до нескольких недель.
, Франция), ВАСТЕС (Becton Dickinson, США), Sensititre TREK Diagnostic Systems (Thermo Scientific, США). С использованием данных систем можно получить количественную информацию об уровне устойчивости микроорганизмов к различным препаратам, однако время проведения анализа для получения данных о профиле резистентности составляет от 7 суток до нескольких недель.
В последние годы основу лабораторной диагностики многих стран мира составляют методы амплификации нуклеиновых кислот. К преимуществам ПЦР-методов относится быстрота, возможность автоматизации и одновременного проведения большого количества исследований, возможность проводить исследования непосредственно с биологическим материалом, минуя стадию культивирования микроорганизма. Однако на чувствительность исследования могут влиять различные ингибирующие факторы, вследствие которых возможны ложноотрицательные результаты. К недостаткам этого метода можно также отнести большую требовательность к чистоте используемых реагентов.
В литературе имеются работы, посвященные одновременной идентификации ряда возбудителей ИПНРФ на основе ПЦР-амплификации с последующей детекцией продуктов методом гибридизации, гель-электрофореза, блоттинга на мембране и др. Описан способ проведения ПЦР для одновременной детекции 7 патогенов: N. gonorrhoeae, С. trachomatis, Т. vaginalis, М. genitalium, Т. pallidum, вирусы герпеса (тип 1 и 2) (Muvunyi С.М., Dhont N., Verhelst R., et al. Evaluation of a new multiplex polymerase chain reaction assay STD finder for the simultaneous detection of 7 sexually transmitted disease pathogens. Diagnostic Microbiol. Infect. Dis. 2011, v. 71 (1), p. 29-37. Описан метод мультиплексной ПЦР и линейного блота для одновременной идентификации 14 микроорганизмов, включающих Т. vaginalis, S. pneumoniae, N. gonorrhoeae, С. trachomatis, U. parvum, U. urealyticum, G. vaginalis, H. influenzae, HSV-1, HSV-2, N. meningitidis, M. hominis, M. genitalium, аденовирусы. Детекцию проводили с использованием хемилюминесцентной реакции. Для каждого организма использовали две специфические пары зондов (McKechnie M.L, Kong F., Gilbert G.L. Simultaneous direct identification of genital microorganisms in voided urine using multiplex PCR-based reverse-line blot assays. Methods Mol. Biol., 2013; v. 943, p. 229-245).
Разработана тест-система на основе мультиплексной ПЦР и реверсивного линейного блота для идентификации 22 факторов вирулентности кишечной палочки, вызывающей развитие негонококкового уретрита (Kudinha Т., Kong F., Johnson J.R., Andrew S.D., Anderson P., Gilbert, G.L. Multiplex PCR-based reverse line blot assay for simultaneous detection of 22 virulence genes in uropathogenic Escherichia coli. Applied and Environmental Microbiology, 2012, v. 78, p. 1198-1202).
Описан ПЦР-метод для одновременного скрининга 13 агентов, ассоциированных с бактериальным вагинозом: G. vaginalis, М. curtisii, М. mulieris, В. fragilis, М. hominis, А. vaginae, U. urealyticum, Megasphaera (тип I), Clostridia, Sneathia sanguinegens, M. genitalium (Malaguti N., Bahls L.D., Uchimura N.S., Gimenes F., Consolaro M.E. Sensitive detection of thirteen bacterial vaginosis-associated agents using multiplex polymerase chain reaction. Biomed. Res. Int., 2015. Epub 2015 May 20, doi: 10.1155/2015/645853).
Разработан ряд коммерческих тест-систем для одновременной диагностики двух и более возбудителей ИПНРФ, например, наборы «Амплисенс» (ООО «ИнтерЛабСервис», Россия), Bio-Rad CT/NG/MG assay (США), Abbott RealTime CT/NG и APTIMA Combo 2 Assay (Abbott, США), Roche Cobas 4800 CT/NG assay (Roche, США), Anyplex II STI-7 assay (TOTEM technology, США). STI multiplex array ('Randox', Ирландия), BD Probetec™ ET - CT/GC (Becton Dickinson, США). Чувствительность определения составляет 500 клеток/мл. Ни одна из существующих коммерческих тест-систем не позволяет одновременно с идентификацией проводить определение генетических детерминант устойчивости к антимикробным препаратам.
Во всех вышеуказанных работах анализ лекарственной резистентности возбудителей не проводился.
Опубликованы работы по применению ПЦР-методов для анализа мутаций, приводящих к лекарственной устойчивости одного возбудителя, например, N. gonorrhoeae (Dona V., Kasraian S., Lupo A., Guilarte Y.N., Hauser C., Furrer H., Unemo M., Low N., Endimiani A. A Multiplex real-time PCR with high resolution melting analysis for the characterization of cntimicrobial resistance in Neisseria gonorrhoeae. J. Clin. Microbiol, 2016 May 25. pii: JCM.03354-15).
Для проведения многопараметрического анализа нуклеиновых кислот предложена технология хМАР, в которой анализ проводится с использованием суспензии флуоресцентно-маркированных микросфер, на поверхности которых расположены олигонуклеотидные зонды. Описано использование микросфер с детекцией Luminex для анализа 18 микроорганизмов-возбудителей урогенитальных инфекций: С. trachomatis, вирусы герпеса (тип 1 и 2), Т. pallidum, Т. vaginalis, N. gonorrhoeae, М. genitalium, М. hominis, М. pneumonia, М. spermatophilum, Ureaplasma urealyticum, U. parvum, A. vaginalis, G. vaginalis, 3 вида Candida и Lactobacillus species (Schmitt M., Depuydt C., Stalpaert M., Pawlita, M. Bead-based multiplex sexually transmitted infection profiling. Journal of Infection, 2014, v. 69, p. 123-133). Балашовым с соавторами (Balashov S., Mordechai E., Adelson M.E., Gygax S.E. Multiplex bead suspension array for screening Neisseria gonorrhoeae antibiotic resistance genetic determinants in noncultured clinical samples. J. Mol. Diagn., 2013, v. 15, p. 116-129) предложено сочетание метода ПЦР и мультиплексного профилирования на микросферах MicroPlex-xTAG (компания Luminex Corp., США) для одновременного детектирования 29 геномных мутаций и 2 плазмидных генов N.gonorrhoeae, служащих генетическими маркерами устойчивости N.gonorrhoeae к 6 антимикробным препаратам (пенициллину, ципрофлоксацину, цефиксиму, тетрациклину, азитромицину и спектиномицину). Тест-система предназначена для работы только с N. gonorrhoeae и неприменима для идентификации других возбудителей ИПНРФ.
Методы таргетного секвенирования с использованием современных систем, например, Ion PGM sequencer (Life technologies, США), MiSeqDx Instrument (Illumina, США) дают возможность идентифицировать образец и одновременно определить генетические детерминанты антибиотикорезистентности, однако чувствительность этих методов недостаточна для анализа клинического материала. Методы все еще отличаются высокой стоимостью, и поэтому в настоящее время не находят массового применения в рутинной лабораторной диагностике возбудителей ИПНРФ.
Современные способы многопараметрического анализа геномных мишеней включают также манипуляции (гибридизацию, амплификацию) с нуклеиновыми кислотами на ДНК-микроматрицах (микрочипах) - массивах закрепленных на твердой фазе олигонуклеотидных зондов, способных специфично связываться с анализируемыми фрагментами геномов. Запатентовано несколько вариантов ДНК-микрочипов для одновременного обнаружения нескольких возбудителей ИПНРФ. ФГБУ «Государственный научный центр дерматовенерологии и косметологии» Минздрава РФ разработан ДНК-чип для одновременной детекции 28 патогенных, непатогенных и условно-патогенных микроорганизмов мочеполовой сферы человека, на котором производится детекция после проведения мультиплексной ПЦР («ДНК-чип для комплексной диагностики инфекций, передаваемых половым путем», патент РФ на полезную модель 117431, приоритет от 05.11.2009; Лихарева В.В., Фриго Н.В., Рахматулина М.Р., Нурутдинова О.С., Шаталова, Ю.А. Разработка ДНК-чипа для комплексной диагностики ИППП. Вестн. дерматол. венерол., 2009, т. 4, с. 49-57). Биочип для одновременной видовой идентификации нескольких инфекционных агентов запатентован ЗАО «Молекулярно-медицинские технологии» (патент РФ №2348695, дата приоритета 23.05.2006, «Дифференцирующий и специфический олигонуклеотиды для идентификации последовательностей ДНК инфекционных агентов в биологических материалах, способ видовой идентификации инфекционных агентов, биочип и набор для осуществления этого способа»).
Имеется патент корейских исследователей на ДНК чип, предназначенный для диагностики инфекций мочеполовых путей (DNA chip for diagnosis of genitourinary infections, заявка на патент США №20140336082). Описан ДНК-микрочип для одновременного аназиза 17 возбудителей заболеваний, передающихся половым путем: N. gonorrhoeae, С. trachomatis, М. genitalium, М. hominis, Ureaplasma, вирусы герпеса 1 и 2 типа, папилломавирусы различных типов (Сао В., Wang S., Tian Z., Hu P., Feng L., Wang L. DNA microarray characterization of pathogens associated with sexually transmitted diseases, PLoS One., 2015, v. 10(7): e0133927). Чип содержал 34 зонда, включающие фрагменты 16S РНК. Вышеописанные ДНК-чипы предназначены только для детекции возбудителей инфекций, но не для идентификации детерминант лекарственной устойчивости.
Опубликован олигонуклеотидный микрочип для детекции точечных мутаций в генах gyrA и parC N. gonorrhoeae, приводящих к лекарственной устойчивости (Zhou W., Du W., et al. Detection of gyrA and parC mutations associated with ciprofloxacin resistance in Neisseria gonorrhoeae by use of oligonucleotide biochip technology. J. Clin. Microbiol., 2004, v. 42 (12), p. 5819-5824). Микрочип позволяет детектировать 15 детерминант, приводящих к резистентности исключительно к ципрофлоксацину, в частности, мутации остатков S91 and D95 белка GyrA, остатков Е91 и S87 белка ParC.
Таким образом, известные к настоящему времени методы молекулярной мультиплексной диагностики возбудителей ИПНРФ решают либо задачи идентификации микроорганизмов, либо определения маркеров резистентности к антимикробным препаратам, как правило, только для одного вида микроорганизма. Существует потребность в разработке молекулярно-генетического инструмента для одновременной идентификации ряда наиболее распространенных возбудителей ИПНРФ, а также молекулярных детерминант их устойчивости к широкому спектру актуальных антимикробных препаратов, таких как антибиотики пенициллинового ряда, цефалоспорины, тетрациклины, фторхинолоны, макролиды, нитроимидазолы, спектиномицин. Использование такого метода позволит проводить персонализированную терапию больных, основанную на рациональном назначении химиопрепаратов, значительно уменьшить количество осложнений и неудач терапии воспалительных заболеваний урогенитального тракта, обусловленных микроорганизмами, устойчивыми к антимикробным препаратам.
Раскрытие изобретения
Изобретением предлагается инструмент для одновременной идентификации ряда наиболее распространенных возбудителей ИПНРФ, а также молекулярных детерминант их устойчивости к широкому спектру актуальных антимикробных препаратов в виде ДНК-чипа с иммобилизованными олигонуклеотидными зондами. Заявляемый ДНК-чип выгодно отличается от известных из уровня техники аналитических инструментов возможностью одновременной идентификации генетических детерминант резистентности Treponema pallidum, Neisseria gonorrhoeae, Chlamydia trachomatis, Mycoplasma genitalium, Mycoplasma hominis, Gardnerella vaginalis, Ureaplasma parvum, Ureaplasma urealyticum, Atopobium vaginae, Enterococcus faecalis, Trichomonas vaginalis, Escherichia coli, Mobiluncus mulieris, Streptococcus anginosus, Fusobacterium nucleatum, Staphylococcus epidermidis, Bacteroides fragilis к антимикробным препаратам, включающим антибиотики пенициллинового ряда, цефалоспорины, тетрациклины, фторхинолоны, макролиды, нитроимидазолы, спектиномицин, что дает возможность установления точного диагноза с учетом полимикробного характера заболевания и назначать эффективную персонифицированную терапию.
В своем первом аспекте данное изобретение обеспечивает ДНК-чип, представляющий собой матрицу элементов, размещенных на подложке из стекла или пластика, причем матрица включает:
- элементы с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 1-33, соответствующими 16S рРНК Т. pallidum, N. gonorrhoeae, С. trachomatis, М. genitalium, М. hominis, G. vaginalis, U. parvum, U. urealyticum, A. vaginae, E. faecalis, T. vaginalis, E.coli, M. mulieris, S. anginosus, F. nucleatum, S. epidermidis, B. fragilis для установления принадлежности анализируемой ДНК к перечисленным выше видам микроорганизмов, являющимися возбудителями инфекций, приводящих к нарушению репродуктивных функций человека;
- элементы с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 34-42, соответствующими последовательностям гена gyrA изолятов N. gonorrhoeae дикого типа, либо последовательностям мутантных вариантов гена gyrA, приводящих к аминокислотным заменам S91F, S91T, D95N, D95G, встречающихся у изолятов N. gonorrhoeae, резистентных к фторхинолонам;
- элементы с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 43-55, соответствующими последовательностям гена gyrA изолятов Е. coli дикого типа, либо последовательностям мутантных вариантов гена gyrA, приводящих к аминокислотным заменам S83L, S83W, S83A, D87N, D87G, D87Y встречающихся у изолятов Е. coli, резистентных к фторхинолонам;
- элементы с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 56-58, соответствующими последовательностям гена gyrA изолятов S. epidermidis дикого типа, либо последовательностям мутантных вариантов гена gyrA, приводящих к аминокислотным заменам S83T, S83F, встречающихся у изолятов S. epidermidis, резистентных к фторхинолонам;
- элементы с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 59-63, соответствующими последовательностям гена gyrA изолятов F. nucleatum дикого типа, либо последовательностям мутантных вариантов гена gyrA, приводящих к аминокислотным заменам S83L, S83T, G87R, встречающихся у изолятов F. nucleatum, резистентных к фторхинолонам;
- элементы с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 64-67, соответствующими последовательностям гена gyrA изолятов Е. faecalis дикого типа, либо последовательностям мутантных вариантов гена gyrA, приводящих к аминокислотным заменам S83T, S83I, S83R, встречающихся у изолятов Е. faecalis, резистентных к фторхинолонам;
- элементы с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID 68-73, соответствующими последовательностям гена gyrA изолятов S. anginosus дикого типа, либо последовательностям мутантных вариантов гена gyrA, приводящих к аминокислотным заменам S83T, S83F, встречающихся у изолятов S. anginosus, резистентных к фторхинолонам;
- элементы с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 74-82, соответствующими последовательностям гена parC изолятов N. gonorrhoeae дикого типа, либо последовательностям мутантных вариантов гена parC, приводящих к аминокислотным заменам S87N, S87R, E91Q, E91G, E91K, Е91А, встречающихся у изолятов N. gonorrhoeae, резистентных к фторхинолонам;
- элементы с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 83-90, соответствующими последовательностям гена parC изолятов М. genitalium дикого типа, либо последовательностям мутантных вариантов гена parC, приводящих к аминокислотным заменам S83N, S83I, S83R, D87N, D87Y, D87V, встречающихся у изолятов М. genitalium, резистентных к фторхинолонам;
- элементы с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 91-95, соответствующими последовательностям гена parC изолятов М. hominis дикого типа, либо последовательностям мутантных вариантов гена parC, приводящих к аминокислотным заменам S83I, S83N, E87K, встречающихся у изолятов М. hominis, резистентных к фторхинолонам;
- элементы с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 96-99, соответствующими последовательностям гена parC изолятов U. urealyticum дикого типа, либо последовательностям мутантных вариантов гена parC, приводящих к аминокислотным заменам D82N, S83L, встречающихся у изолятов U. urealyticum, резистентных к фторхинолонам;
- элементы с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 100-103, соответствующими последовательностям генов ntr4tv и ntr6tv изолятов Т. vaginalis дикого типа, либо последовательностям их мутантных вариантов с нуклеотидными заменами C213G (Y71STOP) в гене ntr4tv, А238Т (K80STOP) в гене ntr6tv, встречающихся у изолятов Т. vaginalis, резистентных к нитроимидазолам;
- элементы с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 104-109, соответствующими последовательностям генов nimB, nimC, nimD, nimE, nimF, nimG, наличие которых приводит к резистентности микроорганизма(ов) к нитроимидазолам;
- элемент с иммобилизованными олигонуклеотидами с нуклеотидной последовательностью SEQ ID NO: 110, соответствующей последовательности плазмидных генов blaSHV-12 blaLEN-17, наличие которых приводит к резистентности микроорганизма(ов) к антибиотикам пенициллинового ряда и цефалоспоринам;
- элемент с иммобилизованными олигонуклеотидами с нуклеотидной последовательностью SEQ ID NO: 111, соответствующей последовательности плазмидного гена blaSHV-97, наличие которого приводит к резистентности микроорганизма(ов) к антибиотикам пенициллинового ряда;
- элементы с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 112-117, соответствующими последовательностям плазмидных генов blaTEM и blaSHV, наличие которых приводит к резистентности микроорганизма(ов) к антибиотикам пенициллинового ряда, либо последовательностям мутантного варианта гена blaTEM с аминокислотными заменами М182Т и G238S, одновременное наличие которых приводит к резистентности микроорганизма(ов) к антибиотикам пенициллинового ряда и цефалоспоринам;
- элементы с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 118-119, соответствующими последовательностям гена penA изолятов N. gonorrhoeae дикого типа, либо последовательностям мутантного варианта гена penA, приводящего к инсерции аспарагиновой кислоты (D) в положение 345, встречающегося у изолятов N. gonorrhoeae, резистентных к антибиотикам пенициллинового ряда;
- элементы с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 120-123, соответствующими последовательностям гена ponA изолятов N. gonorrhoeae дикого типа, либо последовательностям мутантного варианта гена ponA с аминокислотной заменой L421P, встречающейся у изолятов N. gonorrhoeae, резистентных к антибиотикам пенициллинового ряда;
- элементы с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 124-126, соответствующими последовательностям гена rpsJ изолятов N. gonorrhoeae дикого типа, либо последовательностям мутантного варианта гена rpsJ c аминокислотной заменой V57M, V57L, встречающихся у изолятов N. gonorrhoeae, резистентных к тетрациклинам;
- элементы с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 127-128, соответствующими последовательностям генов tetM, наличие которых у Е. faecalis, E. coli, М. genitalium, М. hominis, N. gonorrhoeae, G. vaginalis приводит к резистентности к тетрациклинам;
- элемент с иммобилизованными олигонуклеотидами с нуклеотидной последовательностью SEQ ID NO: 129, соответствующей последовательности гена tetO, наличие которого у изолятов Е. faecalis, S. epidermidis приводит к резистентности к тетрациклинам;
- элементы с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 130-139, соответствующими последовательностям 23S РНК изолятов U. urealyticum, U. hominis, S. anginosus, F. nucleatum, E. faecali, T. pallidum, E. coli, N. gonorrhoeae, C. trachomatis, M. genitalium, M. hominis, S. epidermidis, B. fragilis дикого типа, либо последовательностям мутантных вариантов с заменами G2057A, A2058G, А2058С, A2059G, встречающихся у изолятов перечисленных возбудителей, резистентных к макролидам;
- элементы с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 140-141, соответствующими последовательности гена mefA, наличие которого приводит к резистентности S. anginosus, N. gonorrhoeae, S. epidermidis, В. fragilis к макролидам;
- элементы с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 142-150, соответствующими последовательностям 16S РНК изолятов N. gonorrhoeae дикого типа, либо последовательностям мутантных вариантов с заменами С1192Т, G1064C, встречающихся у изолятов N. gonorrhoeae, резистентных к спектиномицину;
- элементы с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 148, 151-155, соответствующими последовательностям 16S РНК изолятов С. trachomatis, Е. coli, F. nucleatum, A. vaginae, Т. pallidum, U. urealyticum, U. hominis, S. anginosus, S. epidermidis, M. genitalium, M. hominis, M. mulieris, B. fragilis дикого типа, либо последовательностям мутантных вариантов с заменами G1058C, G1064C, встречающихся у изолятов перечисленных возбудителей, резистентных к тетрациклину;
- элементы с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 156-161, соответствующими последовательностям гена помпы эффлюкса mtrR дикого типа, либо последовательностям мутантных вариантов промоторной области гена mtrR, приводящих к делеции аденина А в положении -35 или инсерциям тимидина Т или ТТ в положении -10, встречающихся у изолятов возбудителей, резистентных к антибиотикам пенициллинового ряда, тетрациклинам, макролидам, цефалоспоринам;
- элементы с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 162-163, соответствующими последовательности гена поринового белка porB изолятов N. gonorrhoeae дикого типа, либо последовательности мутантного варианта гена porB, приводящей к аминокислотной замене G120K, встречающегося у изолятов N. gonorrhoeae, резистентных к антибиотикам пенициллинового ряда, цефалоспоринам, тетрациклинам.
Еще одним аспектом данного изобретения является набор олигонуклеотидов, используемый для получения ДНК-чипа для идентификации генетических детерминант резистентности микроорганизмов - возбудителей ИПНРФ: Т. pallidum, N. gonorrhoeae, С. trachomatis, М. genitalium, М. hominis, G. vaginalis, U. parvum, U. urealyticum, A. vaginae, E. faecalis, T. vaginalis, E. coli, M. mulieris, S. anginosus, F. nucleatum, S. epidermidis, B. fragilis, к антимикробным препаратам, включающим антибиотики пенициллинового ряда, цефалоспорины, тетрациклины, фторхинолоны, макролиды, нитроимидазолы, спектиномицин, причем олигонуклеотиды имеют последовательности SEQ ID NO: 1-163.
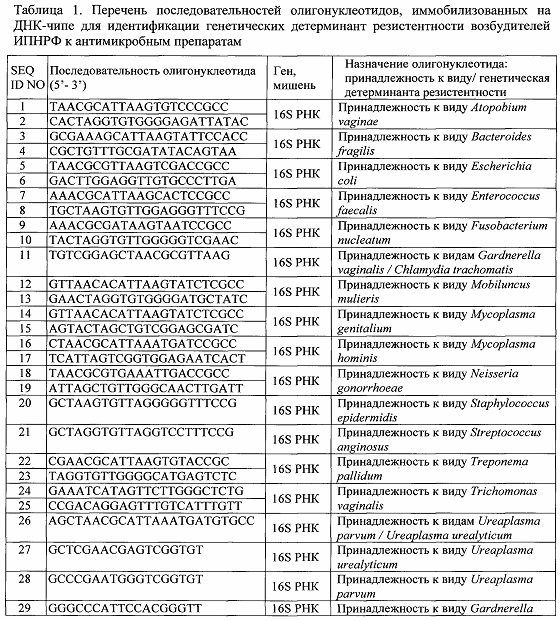
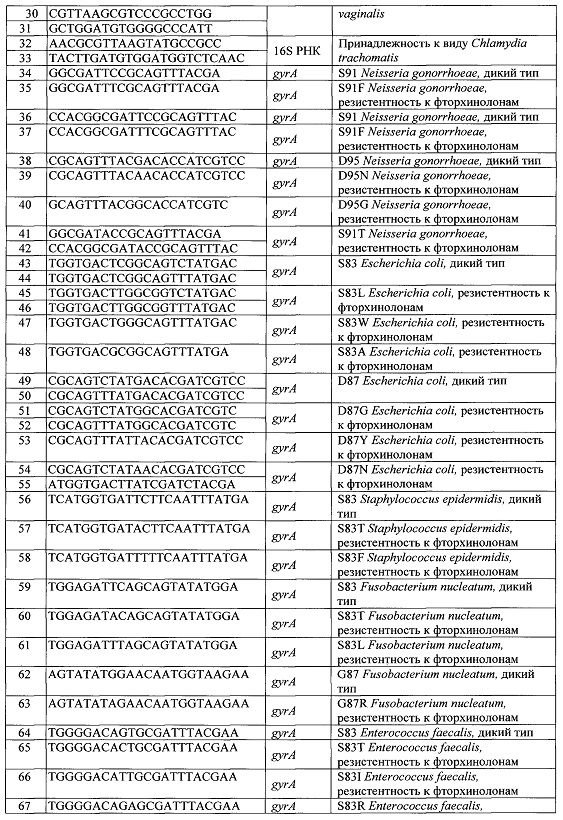
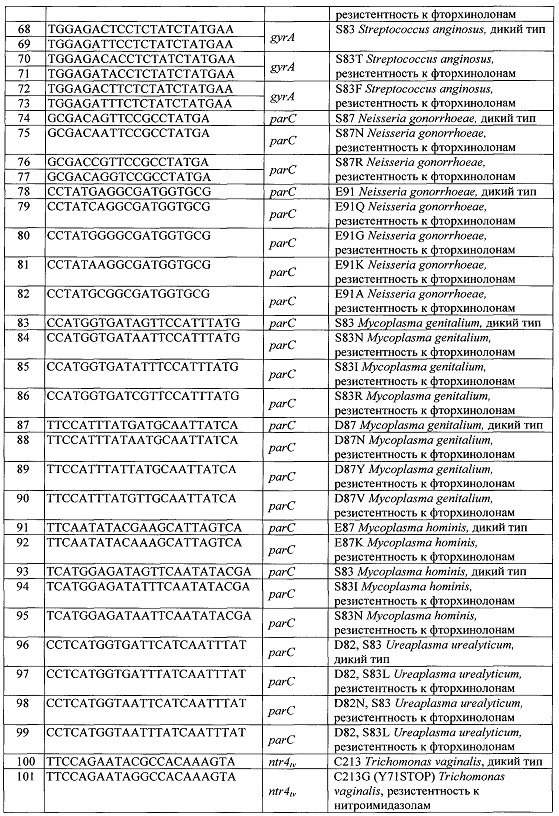
Перечень последовательностей олигонуклеотидов, используемых для изготовления ДНК-чипа, и их SEQ ID приведены в Таблице 1.

Перечень фигур
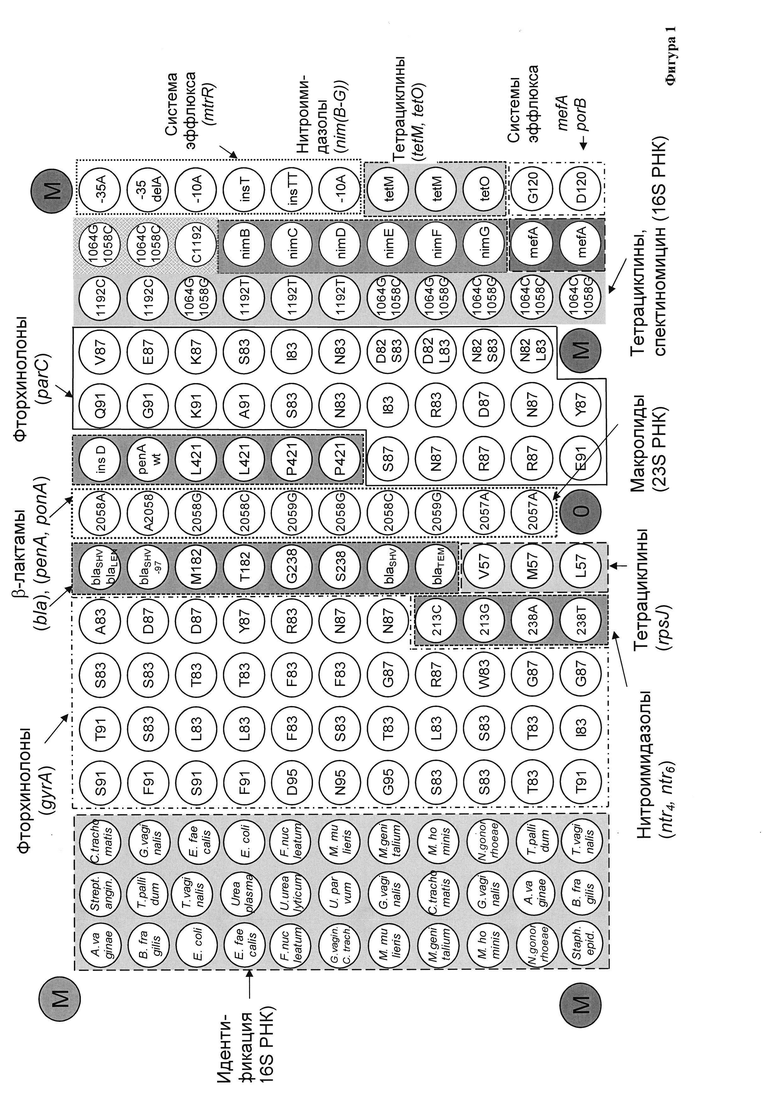
Фигура 1. Схема размещения элементов ДНК-чипа для идентификации генетических детерминант резистентности возбудителей ИПНРФ Т. pallidum, N. gonorrhoeae, С. trachomatis, М. genitalium, М. hominis, G. vaginalis, U. parvum, U. urealyticum, A. vaginae, E. faecalis, T. vaginalis, E. coli, M. mulieris, S. anginosus, F. nucleatum, S. epidermidis, B. fragilis к антимикробным препаратам, включающим антибиотики пенициллинового ряда, цефалоспорины, тетрациклины, фторхинолоны, макролиды, нитроимидазолы, спектиномицин. Элементы ДНК-чипа представлены в виде кружков, надписи внутри которых указывают назначение элементов: анализ принадлежности анализируемой ДНК к виду возбудителя ИПНРФ, анализ вариабельных аминокислот/нуклеотидов на соответствие дикому типу либо мутатному варианту изолята возбудителя ИПНРФ; идентификация маркерного гена резистентности (название гена).
М - элементы, содержащие флуоресцентный маркер;
О - элементы, не содержащие иммобилизованные олигонуклеотиды, которые используются для вычисления фонового сигнала.
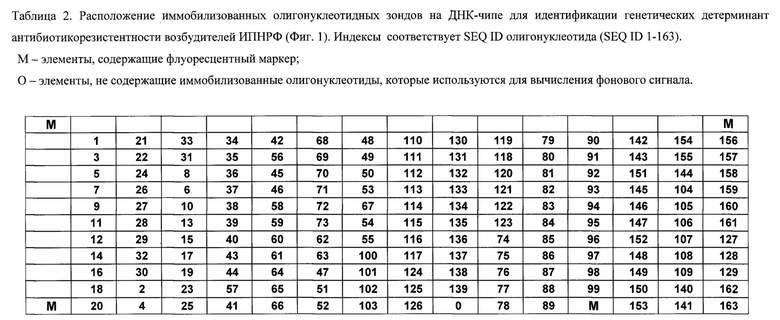
Расположение иммобилизованных на ДНК-чипе олигонуклеотидных зондов в соответствии с SEQ ID приведено в Таблице 2.
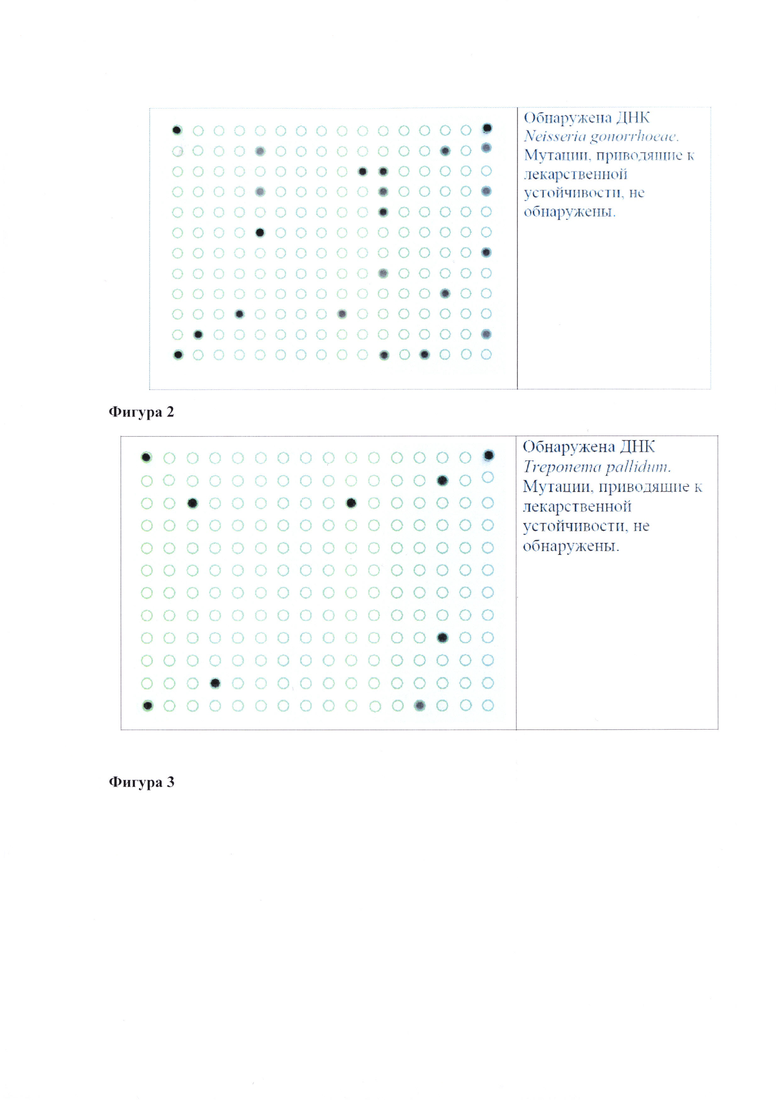
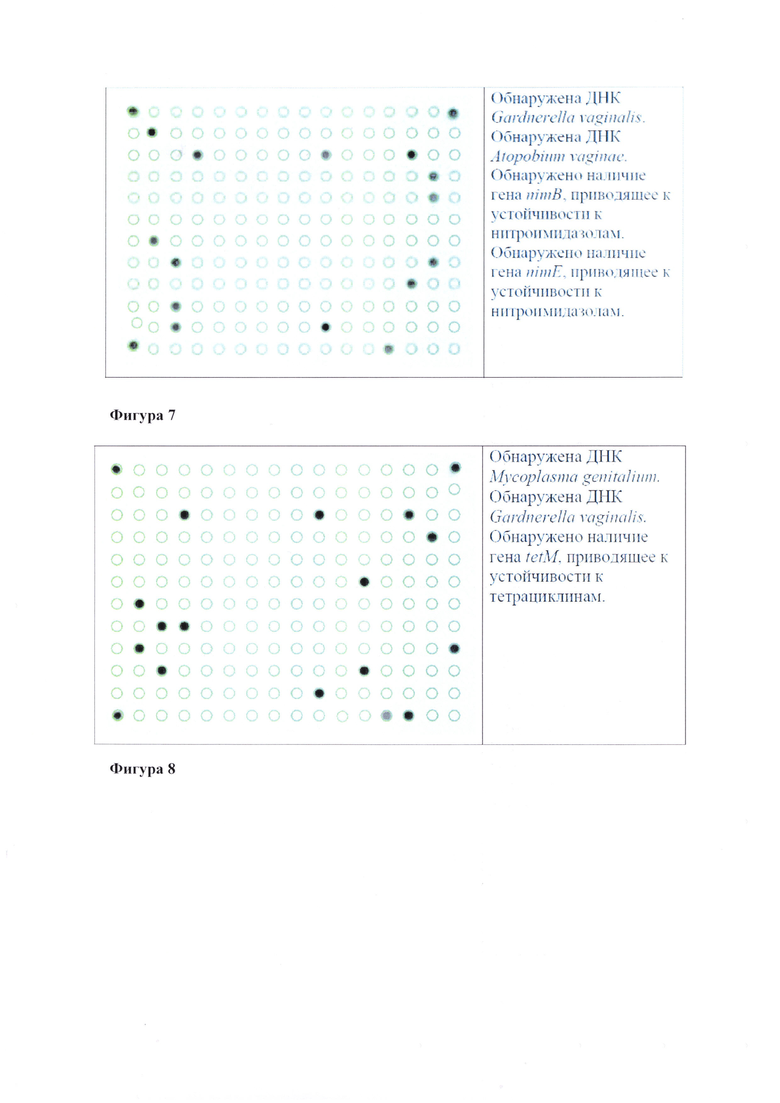
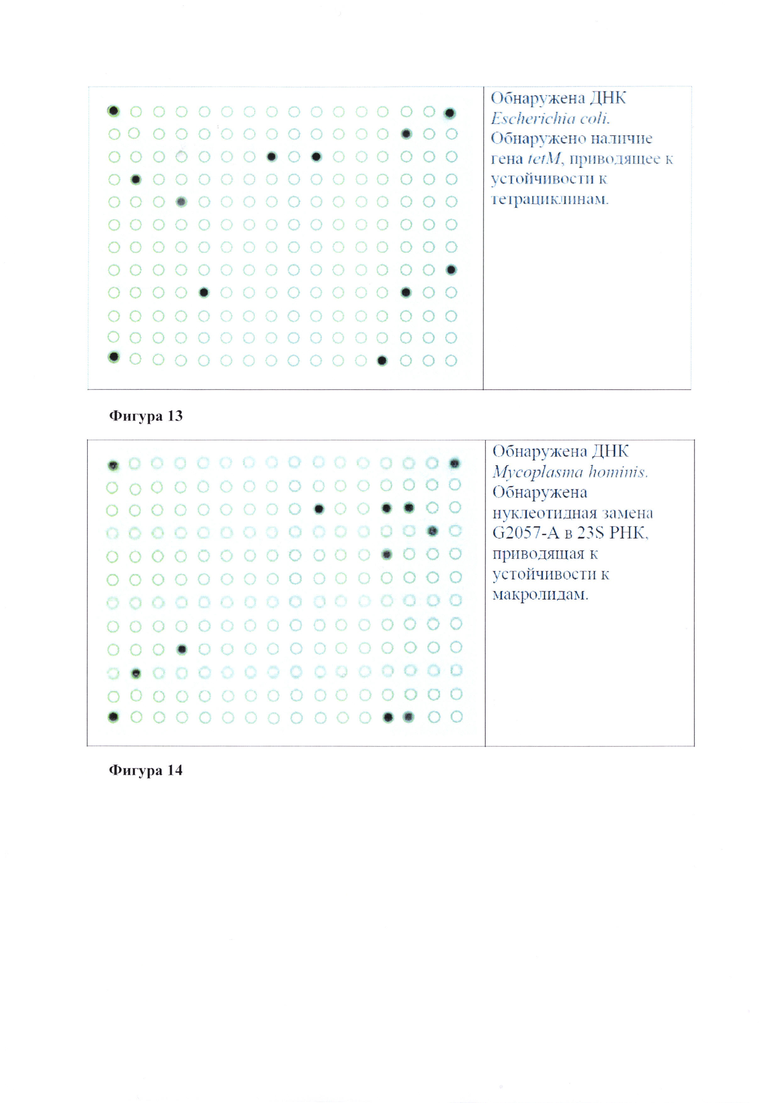
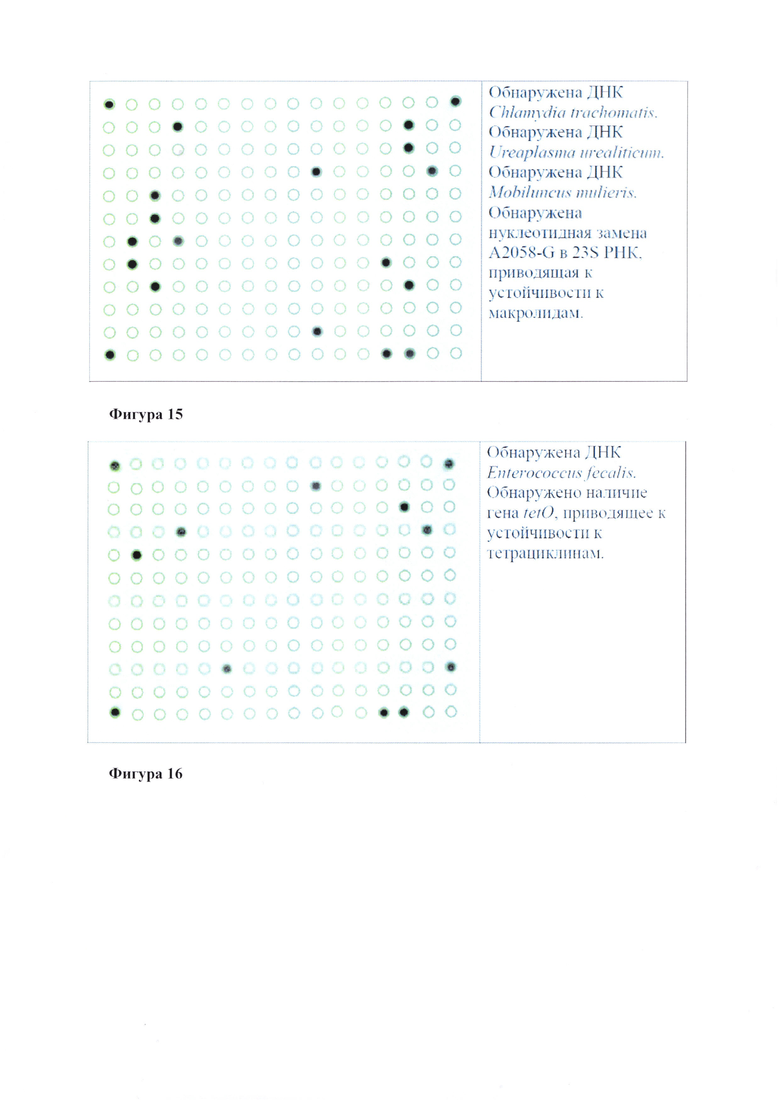
Фигуры 2-22. Гибридизационные картины ДНК-чипов и результаты их интерпретации при анализе образцов ДНК изолятов возбудителей ИПНРФ, различающихся профилями антибиотикорезистентности.
Осуществление изобретения
Целью изобретения являлось конструирование инструмента для определения молекулярных детерминант устойчивости ряда наиболее распространенных возбудителей ИПНРФ к широкому спектру актуальных антимикробных препаратов. В данном изобретении предлагается ДНК-чип - матрица элементов с иммобилизованными олигонуклеотидными зондами для идентификации генетических детерминант резистентности Т. pallidum, N. gonorrhoeae, С. trachomatis, М. genitalium, М. hominis, G. vaginalis, U. parvum, U. urealyticum, A. vaginae, E. faecalis, T. vaginalis, E. coli, M. mulieris, S. anginosus, F. nucleatum, S. epidermidis, B. fragilis, к антимикробным препаратам, включающим антибиотики пенициллинового ряда, цефалоспорины, тетрациклины, фторхинолоны, макролиды, нитроимидазолы, спектиномицин, а также набор олигонуклеотидов, используемый для получения ДНК-чипа.
ДНК-чип представляет собой стеклянную или пластиковую подложку, на которой закреплены олигонуклеотидные зонды. Олигонуклеотиды могут быть адсорбированы на подложке, закреплены (иммобилизованы) на подложке путем образования ковалентных связей, через гидрофобные взаимодействия, комплексообразование и т.д. ДНК-чип может быть получен нанесением водных растворов олигонуклеотидов непосредственно на подложку с получением варианта так называемого «планарного» (двумерного) чипа (например, согласно Matson R.S. Microarray Methods and Protocols. 2009. CRC Press, ISBN 9781420046656), либо представлять собой массив трехмерных гидрогелевых ячеек полусферической формы, содержащих ковалентно иммобилизованные олигонуклеотиды. Трехмерный ДНК-чип может быть изготовлен, например, методом сополимеризационной иммобилизации (патент РФ №2216547, патент ЕР 1437368; Rubina A.Y., Pan'kov S.V. et al., Hydrogel drop microchips with immobilized DNA: properties and methods for large-scale production. Anal. Biochem., 2004, v. 325, p. 92-106). Один из способов получения ДНК-чипа приведен в Примере 1.
Схема расположения олигонуклеотидов на ДНК-чипе представлена на Фиг. 1. Биочип содержит 168 элементов, включая 163 ячейки с иммобилизованными олигонуклеотидами, 1 ячейку, не содержащую зондов, для вычисления фонового сигнала (ячейка с индексом «О»), и 4 ячейки, содержащие иммобилизованный маркер (ячейки с индексом «М»). Ячейки с маркером используются для правильного позиционирования ДНК-чипа при регистрации гибридизационной картины и ее обработки программным обеспечением анализатора биочипов (сканера) при автоматическом получении результатов.
ДНК-чип позволяет устанавливать принадлежность анализируемой ДНК к видам микроорганизмов, являющимися возбудителями инфекций, включающий Т. pallidum, N. gonorrhoeae, С. trachomatis, М. genitalium, М. hominis, G. vaginalis, U. parvum, U. urealyticum, A. vaginae, E. faecalis, T. vaginalis, E. coli, M. mulieris, S. anginosus, F. nucleatum, S. epidermidis, B. fragilis, на основе видоспецифичного полиморфизма в генах 16S рРНК. Для этой цели используются элементы с иммобилизованными олигонуклеотидными зондами с последовательностями, имеющими SEQ ID NO: 1-33.
Выбор анализируемых генетических детерминант резистентности осуществляли с учетом анализа современной литературы, таким образом, чтобы охватить максимально широкий спектр актуальных клинически значимых механизмов формирования устойчивости возбудителей ИПНРФ к антимикробным препаратам (Shaskolskiy В, Dementieva Е, Leinsoo A, Runina A, Vorobyev D, Plakhova X, Kubanov A, Deryabin D, Gryadunov D. 2016. Drug resistance mechanisms in bacteria causing sexually transmitted diseases and associated with vaginosis. Frontiers in Microbology. 7: 747).
Одновременно ДНК-чип позволяет определять:
- мутации в гене gyrA N. gonorrhoeae S91F, S91T, D95N, D95G, встречающиеся у изолятов N. gonorrhoeae, резистентных к фторхинолонам. Используются элементы с иммобилизованными олигонуклеотидными зондами с последовательностями, имеющими SEQ ID NO: 34-42;
- мутации в гене gyrA Е. coli S83L, S83W, S83A, D87N, D87G, D87Y, встречающиеся у изолятов Е. coli, резистентных к фторхинолонам. Используются элементы с иммобилизованными олигонуклеотидными зондами с последовательностями, имеющими SEQ ID NO: 43-55;
- мутации в гене gyrA S. epidermidis S83T, S83F, встречающиеся у изолятов S. epidermidis, резистентных к фторхинолонам. Используются элементы с иммобилизованными олигонуклеотидными зондами с последовательностями, имеющими SEQ ID NO: 56-58;
- мутации в гене gyrA F. nucleatum S83L, S83T, G87R, встречающиеся у изолятов F. nucleatum, резистентных к фторхинолонам. Используются элементы с иммобилизованными олигонуклеотидными зондами с последовательностями, имеющими SEQ ID NO: 59-63;
- мутации в гене gyrA Е. faecalis S83T, S83I, S83R, встречающиеся у изолятов Е. faecalis, резистентных к фторхинолонам. Используются элементы с иммобилизованными олигонуклеотидными зондами с последовательностями, имеющими SEQ ID NO: 64-67;
- мутации в гене gyrA S. anginosus S83T, S83F, встречающиеся у изолятов S. anginosus, резистентных к фторхинолонам. Используются элементы с иммобилизованными олигонуклеотидными зондами с последовательностями, имеющими SEQ ID NO: 68-73;
- мутации в гене parC N. gonorrhoeae S87N, S87R, E91Q, E91G, E91K, Е91А, встречающиеся у изолятов N. gonorrhoeae, резистентных к фторхинолонам. Используются элементы с иммобилизованными олигонуклеотидными зондами с последовательностями, имеющими SEQ ID NO: 74-82;
- мутации в гене parC М. genitalium S83N, S83I, S83R, D87N, D87Y, D87V, встречающиеся у изолятов М. genitalium, резистентных к фторхинолонам. Используются элементы с иммобилизованными олигонуклеотидными зондами с последовательностями, имеющими SEQ ID NO: 83-90;
- мутации в гене parC М. hominis S83I, S83N, E87K, встречающиеся у изолятов М. hominis, резистентных к фторхинолонам. Используются элементы с иммобилизованными олигонуклеотидными зондами с последовательностями, имеющими SEQ ID NO: 91-95;
- мутации в гене parC U. urealyticum D82N, S83L, встречающиеся у изолятов U. urealyticum, резистентных к фторхинолонам. Используются элементы с иммобилизованными олигонуклеотидными зондами с последовательностями, имеющими SEQ ID NO: 96-99;
- мутации в гене ntr4tv C213G (Y71STOP) и гене ntr6tv А238Т (K80STOP) Т. vaginalis, встречающиеся у изолятов Т. vaginalis, резистентных к нитроимидазолам. Используются элементы с иммобилизованными олигонуклеотидными зондами с последовательностями, имеющими SEQ ID NO: 100-103;
- наличие в образце анализируемой ДНК генов nimB, nimC, nimD, nimE, nimF, nimG, ассоциированных с резистентностью микроорганизма(ов) к нитроимидазолам. Используются элементы с иммобилизованными олигонуклеотидными зондами с последовательностями, имеющими SEQ ID NO: 104-109;
- наличие в образце анализируемой ДНК плазмид blaSHV-12 blaLEN-17, характеризующее резистентность микроорганизма(ов) к антибиотикам пенициллинового ряда и цефалоспоринам. Используется элемент с иммобилизованным олигонуклеотидным зондом с последовательностью, имеющей SEQ ID NO: 110;
- наличие в образце анализируемой ДНК плазмиды blaSHV-97, характеризующее резистентность микроорганизма(ов) к антибиотикам пенициллинового ряда. Используется элемент с иммобилизованным олигонуклеотидным зондом с последовательностью, имеющей SEQ ID NO: 111;
- наличие в образце анализируемой ДНК плазмид blaTEM blaSHV, характеризующее резистентность микроорганизма(ов) к антибиотикам пенициллинового ряда, а также мутации в плазмиде blaTEM М182Т и G238S, ассоциированные с резистентностью к антибиотикам пенициллинового ряда и цефалоспоринам. Используются элементы с иммобилизованными олигонуклеотидными зондами с последовательностями, имеющими SEQ ID NO: 112-117;
- мутация в гене penA N. gonorrhoeae: инсерция аспарагиновой кислоты D в положение 345, встречающаяся у изолятов N. gonorrhoeae, резистентных к антибиотикам пенициллинового ряда. Используются элементы с иммобилизованными олигонуклеотидными зондами с последовательностями, имеющими SEQ ID NO: 118-119;
- мутация в гене ponA N. gonorrhea L421P, встречающаяся у изолятов N. gonorrhoeae, резистентных к антибиотикам пенициллинового ряда. Используются элементы с иммобилизованными олигонуклеотидными зондами с последовательностями, имеющими SEQ ID NO: 120-123;
- мутации в гене rpsJ N. gonorrhoeae V57M, V57L, встречающиеся у изолятов N. gonorrhoeae, резистентных к тетрациклинам. Используются элементы с иммобилизованными олигонуклеотидными зондами с последовательностями, имеющими SEQ ID NO: 124-126;
- наличие в образце анализируемой ДНК гена tetM,, встречающегося у изолятов Е. faecalis, E. coli, М. genitalium, М. hominis, N. gonorrhoeae, G. vaginalis, резистентных к тетрациклинам. Используются элементы с иммобилизованными олигонуклеотидными зондами с последовательностями, имеющими SEQ ID NO: 127-128;
- наличие в образце анализируемой ДНК гена tetO, встречающегося у изолятов Е. faecalis, S. epidermidis, резистентных к тетрациклинам. Используется элемент с иммобилизованным олигонуклеотидным зондом с последовательностью, имеющей SEQ ID NO: 129;
- мутации в 23S РНК U. urealyticum, U. hominis, S. anginosus, F. nucleatum, E. faecali, T. pallidum, E. coli, N. gonorrhoeae, C. trachomatis, M. genitalium, M. hominis, S. epidermidis, B. fragilis G2057A, A2058G, A2058C, A2059G, встречающиеся у изолятов перечисленных возбудителей, резистентных к макролидам. Используются элементы с иммобилизованными олигонуклеотидными зондами с последовательностями, имеющими SEQ ID NO: 130-139;
- наличие в образце анализируемой ДНК гена помпы эффлюкса mefA S. anginosus, N. gonorrhoeae, S. epidermidis, В. fragilis, наличие которого приводит к резистентности S. anginosus, N. gonorrhoeae, S. epidermidis, В. fragilis к макролидам. Используются элементы с иммобилизованными олигонуклеотидными зондами с последовательностями, имеющими SEQ ID NO: 140-141;
- мутации в 16S РНК N. gonorrhoeae С1192Т, G1064C, встречающиеся у изолятов N. gonorrhoeae, резистентных к спектиномицину. Используются элементы с иммобилизованными олигонуклеотидными зондами с последовательностями, имеющими SEQ ID NO: 142-150;
- мутации в 16S РНК С. trachomatis, Е. coli, F. nucleatum, A. vaginae, Т. pallidum, U. urealyticum, U. hominis, S. anginosus, S. epidermidis, M. genitalium, M. hominis, M. mulieris, B. fragilis G1058C, G1064C, встречающиеся у изолятов перечисленных возбудителей, резистентных к тетрациклину. Используются элементы с иммобилизованными олигонуклеотидными зондами с последовательностями, имеющими SEQ ID NO: 148, 151-155;
- мутации в гене помпы эффлюкса mtrR: делеция аденина А в положении -35 промоторной области или инсерция тимидина Т или ТТ в положении -10, встречающиеся у изолятов возбудителей, резистентных к антибиотикам пенициллинового ряда, тетрациклинам, макролидам, цефалоспоринам. Используются элементы с иммобилизованными олигонуклеотидными зондами с последовательностями, имеющими SEQ ID NO: 156-161;
- мутация в гене поринового белка porВ N. gonorrhoeae G120K, встречающаясяя у изолятов N. gonorrhoeae, резистентных к антибиотикам пенициллинового ряда, цефалоспоринам, тетрациклинам. Используются элементы с иммобилизованными олигонуклеотидными зондами с последовательностями, имеющими SEQ ID NO: 162-163.
При выборе олигонуклеотидных зондов для иммобилизации на ДНК-чипе учитывают размер и сложность анализируемой последовательности, в частности, наличие повторов и протяженных гомополимерных последовательностей, определяют длину дискриминирующих олигонуклеотидов, обеспечивающую их специфичность в отношении анализируемой последовательности. Для каждой позиции, для которой известны мутации либо видоспецифичный однонуклеотидный полиморфизм, подбирают набор специфичных дискриминирующих олигонуклеотидов, способный выявлять известные варианты замен. С использованием программного обеспечения, например, Oligo v. 6.3 (Molecular Biology Insights Inc., США), рассчитывают температуры плавления олигонуклеотидов и, варьируя их длину, добиваются того, чтобы разброс температур плавления олигонуклеотидов составлял не более 2-3°С. Избегают таких олигонуклеотидов, которые способны формировать вторичные структуры типа шпильки с высокими температурами плавления. Положение определяемых вариабельных нуклеотидов и других нуклеотидных перестроек выбирают по возможности не далее 1-4 нуклеотида от середины соответствующего дискриминирующего олигонуклеотида.
Процедура проведения идентификации генетических детерминант антибиотикорезистентности возбудителей ИПНРФ к антимикробным препаратам с использованием ДНК-чипа включает (а) подготовку клинического образца (материал соскобов со слизистой оболочки органов мочеполовой системы), (б) ПЦР-амплификацию с использованием олигонуклеотидных праймеров, комплементарных соответствующим фрагментам ДНК возбудителей, (в) гибридизацию полученных ПЦР-продуктов на ДНК-чипах, (г) регистрацию и интерпретацию результатов гибридизации.
ПЦР-амплификацию проводят любым способом, позволяющим получить фрагменты ДНК, соответствующие анализируемым последовательностям, например, как описано в опубликованных ранее работах: Зименков Д.В., Кулагина Е.В., Антонова О.В., Суржиков С.А., Беспятых Ю.А., Шитиков Е.А., Ильина Е.Н., Михайлович В.М., Заседателев А.С. Грядунов Д.А. Анализ генетических детерминант множественной и широкой лекарственной резистентности возбудителя туберкулеза с использованием олигонуклеотидного микрочипа. Молекулярная биология, 2014, т. 48(2), с. 251-264; Грядунов Д.А, Гетман И.А., Чижова С.Н., Михайлович В.М., Заседателев А.С., Романов Г.А. Идентификация генно-модифицированных источников растительного происхождения в пищевых продуктах и сырье с использованием гидрогелевого олигонуклеотидного микрочипа. Молекулярная биология, 2011, т. 45(6), с. 973-983; Zimenkov DV, Kulagina EV, Antonova OV, Zhuravlev VY, Gryadunov DA. 2016. Simultaneous drug resistance detection and genotyping of Mycobacterium tuberculosis using a low-density hydrogel microarray. Journal of Antimicrobial Chemotherapy. 71 (6): 1520-1531.
Гибридизацию полученных ПЦР-продуктов на ДНК-чипе проводят в растворе, содержащем буферный компонент для поддержания рН, соль для создания ионной силы и хаотропный (дестабилизирующий водородные связи) агент, в герметичной гибридизационной камере при температуре, зависящей от температуры плавления иммобилизованных на микрочипе дискриминирующих олигонуклеотидов, как описано в работе Mikhailovich V.M., Lapa S.A., Gryadunov D.A., et al. Detection of rifampicin-resistant Mycobacterium tuberculosis strains by hybridization and polymerase chain reaction on a specialized ТВ-Microchip. Journal of Clinical Microbiology, 2001, v. 131(1), p. 94-98.
После проведения гибридизации проводят регистрацию гибридизационных картин и их интепретацию, устанавливая принадлежность анализируемой ДНК к видам микроорганизмов, являющихся возбудителями ИПНРФ и идентифицируя генетические детерминанты резистентности к антимикробным препаратам, включающим антибиотики пенициллинового ряда, цефалоспорины, тетрациклины, фторхинолоны, макролиды, нитроимидазолы, спектиномицин.
Принадлежность анализируемой ДНК к виду микроорганизма - возбудителя ИПНРФ устанавливают, анализируя сигналы ячеек ДНК-чипа с иммобилизованными олигонуклеотидами с SEQ ID NO: 1-33 (Фиг. 1, Таблица 2) и сравнивая его со значением сигнала в элементе ДНК-чипа, не содержащего олигонуклеотидов I0 (сигнал ячейки с индексом «О»). Если для максимального сигнала в ячейках A.vaginae (SEQ ID NO: 1, 2) выполняется соотношение Imax/I0≥2, то делают вывод о наличии в образце ДНК Atopobium vaginae; если это соотношение выполняется для ячеек В. fragilis (SEQ ID NO: 3, 4), то делают вывод о наличии в образце ДНК Bacteroides fragilis, для ячеек E. coli (SEQ ID NO: 5, 6) - ДНК Escherichia coli и т.д. Если для возбудителя отношение Imax/I0<2, то делают вывод об отсутствии ДНК данного возбудителя в анализируемом образце, и далее анализ его антибиотикорезистентности не проводят.
Анализ элементов, содержащих зонды, специфичные для мутаций, приводящих к аминокислотым/нуклеотидным заменам, проводят посредством сравнения интенсивности сигналов элементов, содержащих олигонуклеотиды, соответствующие дикому типу и мутантным вариантам. Если максимальный сигнал регистрируют в элементе, соответствующем ДНК возбудителя дикого типа, то это означает, что в данной аминокислотной/нуклеотидной позиции анализируемый образец ДНК не имеет мутаций. Если регистрируют максимальный сигнал в ячейке, соответствующей ДНК с мутацией, это означает, что по данной аминокислотной/нуклеотидной позиции анализируемый образец ДНК несет мутацию, ассоциированную с резистентностью возбудителя к антимикробному препарату (препаратам).
Наличие генетических детерминат резистентности nimB, nimC, nimD, nimE, nimF, nimG, tetM, tetO, blaTEM, blaLEN, blaSHV, mefA определяют путем сравнения сигнала соответствующей ячейки ДНК-чипа с сигналом в элементе ДНК-чипа, не содержащего олигонуклеотидов I0. Например, если выполняется соотношение InimB/I0≥2, считают, что анализируемый образец ДНК содержит ген nimB, ассоциированный с резистентностью к нитроимидазолам; если выполняется соотношение ImefA/I0≥2, считают, что анализируемый образец ДНК содержит ген mefA, ассоциированный с резистентностью S. anginosus, N. gonorrhoeae, S. epidermidis, В. fragilis к макролидам и т.д.
Ниже представлен алгоритм интерпретации результатов, полученных на ДНК-чипе: идентификация генетических детерминант резистентности возбудителей ИПНРФ к антимикробным препаратам.
Элементы ДНК-чипа для анализа резистентности к фторхинолонам (gyrA) (Фиг. 1, Таблица 2):
- максимальный сигнал от ячеек S91, D95 (SEQ ID NO: 34, 36) свидетельствует об отсутствии мутаций в фрагменте гена gyrA N. gonorrhoeae; максимальный сигнал от ячеек F91, Т91, N95, G95 (SEQ ID NO: 35, 37-42) указывает на наличие в данном гене мутаций S91F, S91T, D95N, D95G, ассоциированных с резистентностью N. gonorrhoeae к фторхинолонам;
- максимальный сигнал от ячеек S83, D87 (SEQ ID NO: 43, 44) свидетельствует об отсутствии мутаций в фрагменте гена gyrA Е. coli; максимальный сигнал от ячеек L83, W83, А83, N87, G87, Y87 (SEQ ID NO: 47-55) указывает на наличие в данном гене мутаций S83L, S83W, S83A, D87N, D87G, D87Y, ассоциированных с резистентностью Е. coli к фторхинолонам;
- максимальный сигнал от ячейки S83 (SEQ ID NO: 56) свидетельствует об отсутствии мутаций в фрагменте гена gyrA S. epidermidis; максимальный сигнал от ячеек S83, Т83 (SEQ ID NO: 57, 58) указывает на наличие в данном гене мутаций S83T, S83F, ассоциированных с резистентностью S. epidermidis к фторхинолонам;
- максимальный сигнал от ячеек S83, G87 (SEQ ID NO: 59, 62) свидетельствует об отсутствии мутаций в фрагменте гена gyrA F. nucleatum; максимальный сигнал от ячеек L83, Т83, R87 (SEQ ID NO: 60, 61, 63) указывает на наличие в данном гене мутаций S83L, S83T, G87R, ассоциированных с резистентностью F. nucleatum к фторхинолонам;
- максимальный сигнал от ячейки S83 (SEQ ID NO: 64) свидетельствует об отсутствии мутаций в фрагменте гена gyrA Е. faecalis; максимальный сигнал от ячеек Т83, I83, R83 (SEQ ID NO: 65-67) указывает на наличие в данном гене мутаций S83T, S83I, S83R, ассоциированных с резистентностью Е. faecalis к фторхинолонам;
- максимальный сигнал от ячеек S83 (SEQ ID NO: 68, 69) свидетельствует об отсутствии мутаций в фрагменте гена gyrA S. anginosus; максимальный сигнал от ячеек Т83, F83 (SEQ ID NO: 70-73) указывает на наличие в данном гене мутаций S83T, S83F, ассоциированных с резистентностью S. anginosus к фторхинолонам.
Элементы ДНК-чипа для анализа резистентности к фторхинолонам (parC (Фиг. 1, Таблица 2):
- максимальный сигнал от ячеек S87, Е91 (SEQ ID NO: 74, 78) свидетельствует об отсутствии мутаций в фрагменте гена parC N. gonorrhoeae; максимальный сигнал от ячеек N87, R87, Q91, G91, K91, А91 (SEQ ID NO: 75-77, 79-82) указывает на наличие в данном гене мутаций S87N, S87R, E91Q, E91G, E91K, Е91А, ассоциированных с резистентностью N. gonorrhoeae к фторхинолонам;
- максимальный сигнал от ячеек S83, D87 (SEQ ID NO: 83, 87) свидетельствует об отсутствии мутаций в фрагменте гена parC М. genitalium; максимальный сигнал от ячеек N83, I83, R83, N87, Y87, V87 (SEQ ID NO: 84-86, 88-90) указывает на наличие в данном гене мутаций S83N, S83I, S83R, D87N, D87Y, D87V, ассоциированных с резистентностью М. genitalium к фторхинолонам;
- максимальный сигнал от ячеек S83, Е87 (SEQ ID NO: 91, 93) свидетельствует об отсутствии мутаций в фрагменте гена parC М. hominis; максимальный сигнал от ячеек I83, N83, K87 (SEQ ID NO: 92, 94, 95) указывает на наличие в данном гене мутаций S83I, 83N, E87K, ассоциированных с резистентностью М. hominis к фторхинолонам;
- максимальный сигнал от ячейки D82 S83 (SEQ ID NO: 96) свидетельствует об отсутствии мутаций в фрагменте гена parC U. urealyticum; максимальный сигнал от ячеек N82, L83 (SEQ ID NO: 97-99) указывает на наличие в данном гене мутаций D82N, S83L, ассоциированных с резистентностью U. urealyticum к фторхинолонам;
Элементы ДНК-чипа для анализа резистентности к нитроимидазолам (ntr4tv, ntr6tv) (Фиг. 1, Таблица 2):
- максимальный сигнал от ячеек 213С, 238А (SEQ ID NO: 100, 102) свидетельствует об отсутствии мутаций в генах ntr4tv и ntr6tv, Т. vaginalis; максимальный сигнал от ячеек 213G, 238Т (SEQ ID NO: 101, 103) указывает на наличие в данном гене мутаций C213G (Y71STOP) в гене ntr4tv и А238Т (K80STOP) в гене ntr6tv, встречающихся у изолятов Т. vaginalis, резистентных к нитроимидазолам.
Элементы ДНК-чипа для анализа резистентности к нитроимидазолам (nim(B-G)) (Фиг. 1, Таблица 2):
- анализируют сигналы от ячеек nimB, nimC, nimD, nimE, nimF, nimG (SEQ ID NO: 104-109), и при выполнении соотношения Inim/I0≥2 считают, что анализируемый образец ДНК содержит соответствующий ген nim, ассоциированный с резистентностью к нитроимидазолам.
Элементы ДНК-чипа для анализа резистентности к β-лактамам (bla) (Фиг. 1, Таблица 2):
- анализируют сигналы от ячейки blaSHV-12 blaLEN-17 (SEQ ID NO: 110), и при выполнении соотношения Ibla/I0≥2 считают, что анализируемый образец ДНК содержит ген blaSHV-12 и/или blaLEN-17, ассоциированный с резистентностью к антибиотикам пенициллинового ряда и цефалоспоринам;
- анализируют сигналы от ячейки blaSHV-97 (SEQ ID NO: 111), и при выполнении соотношения Ibla/I0≥2 считают, что анализируемый образец ДНК содержит ген blaSHV-97, ассоциированный с резистентностью к антибиотикам пенициллинового ряда;
- анализируют сигналы от ячеек blaTEM, blaSHV, M182, Т182, G238, S283 (SEQ ID NO: 112-117), и при выполнении соотношения Ibla/I0≥2 считают, что анализируемый образец ДНК содержит соответствующий ген, ассоциированный с резистентностью к антибиотикам пенициллинового ряда; при этом максимальный сигнал от ячеек Т182 и/или S283 указывает на наличие замен М182Т и/или G238S, свидетельствующих о резистентности к антибиотикам пенициллинового ряда и цефалоспоринам
Элементы ДНК-чипа для анализа резистентности к β-лактамам (penA, ponA) (Фиг. 1, Таблица 2):
- максимальный сигнал от ячейки penA wt (SEQ ID NO: 118) свидетельствует об отсутствии мутаций в фрагменте гена penA N. gonorrhoeae; максимальный сигнал от ячейки insD (SEQ ID NO: 119) указывает на наличие в данном гене инсерции аспарагиновой кислоты (D) в положение 345, встречающейся у изолятов N. gonorrhoeae, резистентных к антибиотикам пенициллинового ряда;
- максимальный сигнал от ячеек L421 (SEQ ID NO: 120, 121) свидетельствует об отсутствии мутаций в фрагменте гена ponA N. gonorrhoeae; максимальный сигнал от ячейки Р421 (SEQ ID NO: 122, 123) указывает на наличие в данном гене мутации L421P, ассоциированной с резистентностью N. gonorrhoeae к антибиотикам пенициллинового ряда.
Элементы ДНК-чипа для анализа резистентности к тетрациклинам (rpsJ) (Фиг. 1, Таблица 2):
- максимальный сигнал от ячейки V57 (SEQ ID NO: 124) свидетельствует об отсутствии мутаций в фрагменте гена rpsJ N. gonorrhoeae; максимальный сигнал от ячейки М57, L57 (SEQ ID NO: 125, 126) указывает на наличие в данном гене мутаций V57M, V57L ассоциированных с резистентностью N. gonorrhoeae к тетрациклинам.
Элементы ДНК-чипа для анализа резистентности к тетрациклинам (tetM, tetO) (Фиг. 1, Таблица 2):
- анализируют сигналы от ячеек tetM, tetO (SEQ ID NO: 127-129), и при выполнении соотношения Itet/I0≥2 считают, что анализируемый образец ДНК содержит соответствующий ген, ассоциированный с резистентностью к тетрациклинам (tetM у Е. faecalis, E. coli, М. genitalium, М. hominis, N. gonorrhoeae, tetO у изолятов Е. faecalis, S. epidermidis).
Элементы ДНК-чипа для анализа резистентности к макролидам (23S РНК) (Фиг. 1, Таблица 2):
- максимальный сигнал от ячейки 2058А (SEQ ID NO: 130) свидетельствует об отсутствии мутаций в поз. 257-259 23S РНК U. urealyticum, U. hominis, S. anginosus, F. nucleatum, E. faecalis; максимальный сигнал от ячеек 2058G, 2058С, 2059G (SEQ ID NO: 132-134, 138) указывает на наличие замен A2058G, А2058С, A2059G, G2057A, встречающихся у изолятов перечисленных возбудителей, резистентных к макролидам;
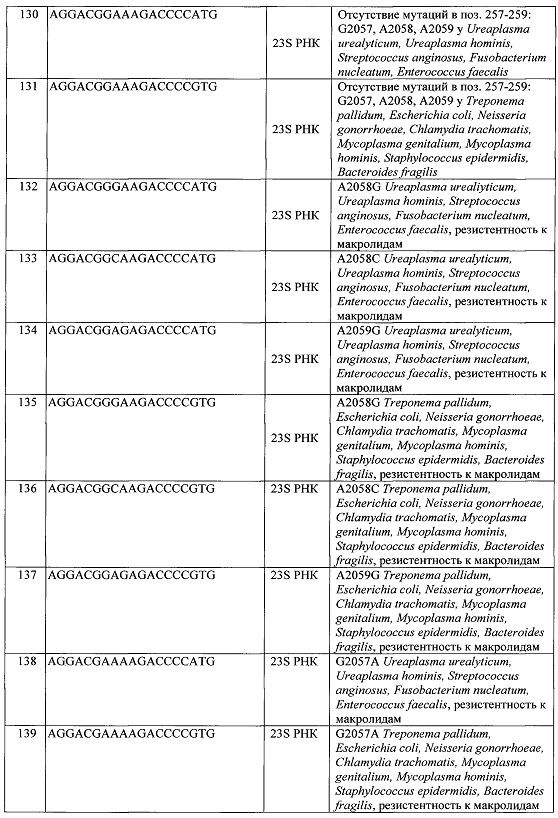
- максимальный сигнал от ячейки А2058 (SEQ ID NO: 131) свидетельствует об отсутствии мутаций в поз. 257-259 23S РНК Т. pallidum, E. coli, N. gonorrhoeae, С. trachomatis, М. genitalium, М. hominis, S. epidermidis, В. fragilis; максимальный сигнал от ячеек 2058G, 2058С, 2059G, 2057А (SEQ ID NO: 135-137, 139) указывает на наличие замен A2058G, А2058С, A2059G, G2057A, встречающихся у изолятов перечисленных возбудителей, резистентных к макролидам.
Элементы ДНК-чипа для анализа резистентности к макролидам (mef) (Фиг. 1, Таблица 2):
- анализируют сигналы от ячеек mefA (SEQ ID NO: 140, 141), и при выполнении соотношения Imef/I0≥2 считают, что анализируемый образец ДНК содержит ген mefA, ассоциированный с резистентностью S. anginosus, N. gonorrhoeae, S. epidermidis, В. fragilis к макролидам.
Элементы ДНК-чипа для анализа резистентности к тетрациклинам, спектиномицину (16S РНК) (Фиг. 1. Таблица 2):
- максимальный сигнал от ячеек 1192С, 1064G-1058G (SEQ ID NO: 142-144, 148) свидетельствует об отсутствии мутаций в 16S РНК N. gonorrhoeae; максимальный сигнал от ячеек 1064C-1058G, 1064С-1058С (SEQ ID NO: 145-147, 149-150) указывает на наличие нуклеотидных замен С1192Т, G1064C, ассоциированных с резистентностью N. gonorrhoeae к спектиномицину.
- максимальный сигнал от ячеек 1064G-1058G (SEQ ID NO: 148, 151) свидетельствует об отсутствии мутаций в 16S РНК С. trachomatis, Е. coli, F. nucleatum, A. vaginae, Т. pallidum, U. urealyticum, U. hominis, S. anginosus, S. epidermidis, M. genitalium, M. hominis, M. mulieris, B. fragilis; максимальный сигнал от ячеек 1064G-1058C, 1064С-1058G, 1064С-1058С (SEQ ID NO: 152-155) указывает на наличие нуклеотидных замен G1058C, G1064C встречающихся у изолятов перечисленных возбудителей, резистентных к тетрациклину.
Элементы ДНК-чипа для анализа гена помпы эффлюкса mtrR (Фиг. 1, Таблица 2):
- максимальный сигнал от ячеек -35А, -10А (SEQ ID NO: 156, 158, 161) свидетельствует об отсутствии мутаций в гене помпы эффлюкса mtrR; максимальный сигнал ячеек -35 delA,
insT, insTT (SEQ ID NO: 157, 159-160) указывает на наличие мутаций в гене mtrR, ассоциированных с резистентностью к антибиотикам пенициллинового ряда, тетрациклинам, макролидам, цефалоспоринам.
Элементы ДНК-чипа для анализа гена поринового белка porВ (Фиг. 1, Таблица 2):
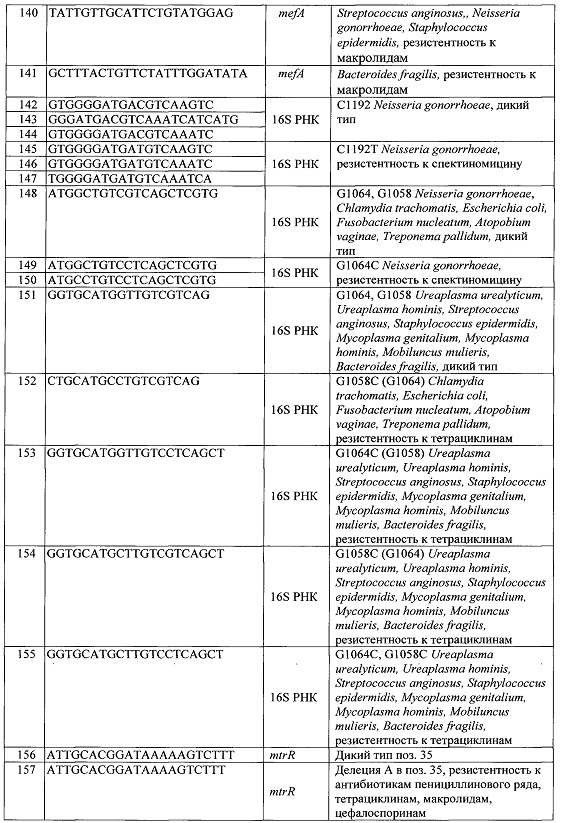
- максимальный сигнал от ячейки G120 (SEQ ID NO: 162) свидетельствует об отсутствии мутаций в гене porВ N. gonorrhoeae; максимальный сигнал ячейки D120 указывает на наличие мутации G120K, встречающейся у изолятов N. gonorrhoeae, резистентных к антибиотикам пенициллинового ряда, цефалоспоринам, тетрациклинам.
Описанный алгоритм анализа может быть реализован в программном обеспечении, позволяющем проводить автоматическую регистрацию и интерпретацию результатов анализа с использованием ДНК-чипа.
Примеры
Пример 1. Изготовление ДНК-чипа для идентификации генетических детерминант антибиотикорезистентности возбудителей инфекций, приводящих к нарушению репродуктивных функций человека
Синтез олигонуклеотидов с последовательностями SEQ ID NO: 1-163 (Таблица 1) для иммобилизации на ДНК-чипе проводили на автоматическом синтезаторе ABI-394 DNA/RNA synthesizer («Applied Biosystems», США) с использованием стандартного фосфорамидитного метода и очищали методом обращенно-фазовой ВЭЖХ (комплекс «Gilson», Франция). В процессе синтеза в олигонуклеотиды вводили спейсер со свободной аминогруппой с использованием 5'-Amino-Modifier С6 («Glen Research», США). ДНК-чипы изготавливали в соответствии с процедурой, описанной ранее (Rubina A.Y., Pan'kov S.V. et al. Hydrogel drop microchips with immobilized DNA: properties and methods for large-scale production. Anal. Biochem., 2004, v. 325, p. 92-106).
Полимеризационные смеси, содержащие смесь гелеобразующих мономеров (производные метакриламида, метилен-бис-акриламида, стабилизаторы и др.) и иммобилизуемые соединения (олигонуклеотиды или флуоресцентный краситель IMD 504), помещали в ячейки 384-луночного планшета. С использованием робота QArray (Genetix, Великобритания) смеси из лунок планшета переносили в виде микрокапель на поверхность пластиковых подложек. Полимеризация геля с одновременной ковалентной иммобилизацией олигонуклеотидов в структуре геля происходила под действием УФ-излучения (УФ-лампа Sylvania GTE F15T8350 BL, Великобритания), 40 мин, 20°С, в токе азота. После полимеризации подложки с массивами гидрогелевых ячеек (микрокапель) промывали 0,1 М фосфатным буфером, водой и высушивали в беспылевой атмосфере.
Схема изготовленного ДНК-чипа на основе гидрогеля для идентификации генетических детерминант антибиотикорезистентности возбудителей ИПНРФ, приведена на Фиг. 1. Расположение иммобилизованных на ДНК-чипе олигонуклеотидных зондов в соответствии с SEQ ID приведено в Таблице 2.
Пример 2. Идентификация генетических детерминант резистентности возбудителей ИПНРФ к антимикробным препаратам с использованием ДНК-чипа
Процедура анализа включала следующие стадии: выделение ДНК из клинического материала, мультиплексную ПЦР использованием набора специфичных олигонуклеотидных праймеров и смеси четырех дезоксинуклеозидтрифосфатов, один из которых содержал флуоресцентную метку, с получением флуоресцентно-меченных фрагментов ДНК; гибридизацию амплифицированных флуоресцентно-меченных продуктов на ДНК-чипах; регистрацию флуоресцентных изображений (гибридизационных картин) и интерпретацию результатов гибридизации.
а) Обработка клинического материала и выделение ДНК
Обработку образцов клинического материала - соскобов со слизистой оболочки органов мочеполовой системы, и выделение тотальной ДНК проводили с использованием набора «ДНК-Экспресс для выделения ДНК из биопроб» (ЗАО «Литех», Россия) согласно рекомендациям производителя.
б) Подготовка праймеров для мультиплексной ПЦР
При выборе последовательностей праймеров для проведения ПЦР использовали базы данных нуклеотидных последовательностей, известные специалистам в данной области, например, база данных http://www.ncbi.nlm.nih.gov/Genbank/index.html. Специфичность праймеров проверяли с помощью программного обеспечения, использующего поиск в базах нуклеотидных последовательностей по алгоритму BLAST (www.ncbi.nlm.nih.gov/BLAST). Перечень праймеров, использованных в данном примере, приведен в Таблице 3. Праймеры синтезировали на автоматическом синтезаторе ABI-394 DNA/RNA synthesizer («Applied Biosystems», США) с использованием стандартного фосфорамидитного метода и очищали методом обращенно-фазовой ВЭЖХ (комплекс «Gilson», Франция).
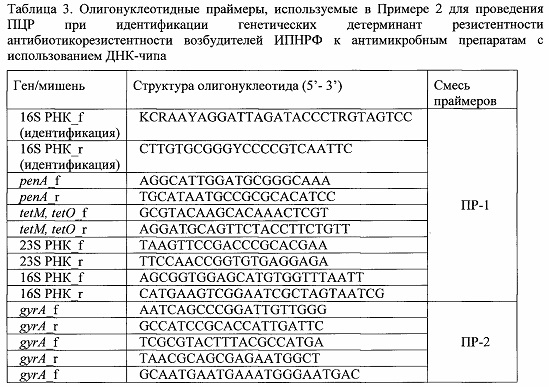
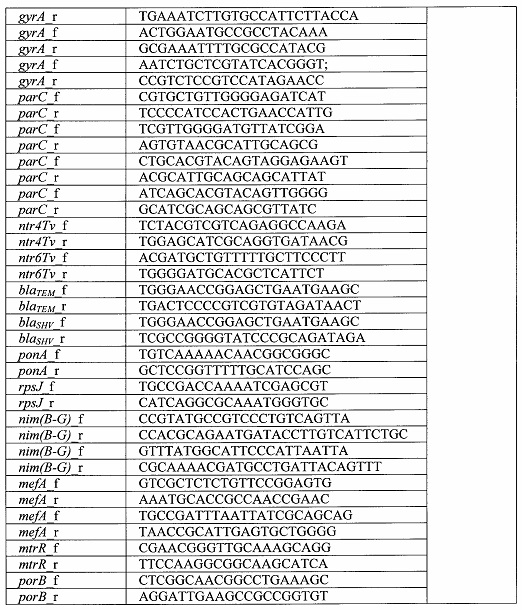
Обозначения: f - прямой праймер, r - обратный праймер,
R=G или A; Y=Т или С; K=G или Т.
в) Проведение мультиплексной ПЦР
Готовили две смеси праймеров для проведения ПЦР ПР-1 и ПР-2, содержащие олигонуклеотиды, указанные в Таблице 3. Количество каждого праймера в реакционной смеси составляет: для смеси ПР-1 - 10 нмоль (прямой праймер), 50 нмоль (обратный праймер), для смеси ПР-2 - 40 нмоль (прямой праймер), 200 нмоль (обратный праймер), Готовили две реакционные смеси для проведения ПЦР, внося в пробирку следующие реагенты (из расчета на 1 реакцию).
Смесь 1:
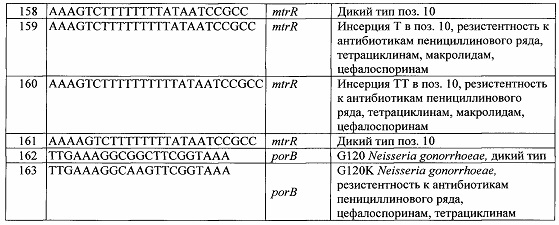
Смесь 2:
В качестве флуоресцентно-меченного субстрата использовали конъюгат дУТФ с флуоресцентным красителем (максимум поглощения при 645 нм, максимум флуоресценции при 670 нм) (Kuznetsova V.E., Spitsyn М.А., Shershov V.E., Guseinov Т.О., Fesenko E.E., Lapa S.A., Ikonnikova A.Iu., Avdonina M.A., Nasedkina T.V., Zasedatelev A.S., Chudinov A.V. Novel fluorescently labeled nucleotides: synthesis, spectral properties and application in polymerase chain reaction. Mendeleev Communications, 2016, v. 26(2), p. 95-98). Флуоресцентный субстрат встраивается ДНК-полимеразой в растущую цепь ДНК в ходе ПЦР.
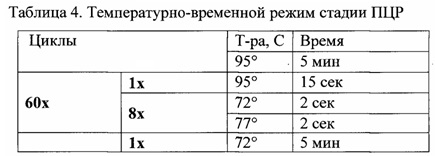
К полученным ПЦР-смесям добавляли по 1 мкл раствора ДНК, полученного из клинического образца. Для отрицательного контрольного образца использовали 1 мкл деионизованной воды. Мультиплексную ПЦР проводили на амплификаторе S1000 (Bio-Rad, США, используя приведенный ниже температурно-временной режим (Таблица 4).
Растворы после проведения ПЦР использовали для проведения гибридизации на ДНК-чипе, изготовленном, как описано в Примере 1.
г) Гибридизация флуоресцентно-меченных продуктов на ДНК-чипах
В пробирку вносили 10 мкл буфера для гибридизации (0,3 М HEPES, рН 7,5, 3,0 М гуанидинтиоцианата, 30 мМ ЭДТА) и по 10 мкл реакционных смесей после проведения ПЦР. 30 мкл полученной гибридизационной смеси помещали в реакционную камеру ДНК-чипа и проводили гибридизацию при 37°С в течение 6-12 часов. По окончании гибридизации ДНК-чип трижды промывали дистиллированной водой при 37°С, снимали гибридизационную камеру, и высушивали ДНК-чип в потоке воздуха,
д) Регистрация и интерпретация результатов гибридизации на ДНК-чипах
Регистрацию флуоресцентных изображений ДНК-чипов (гибридизационных картин) выполняли на универсальном аппаратно-программном комплексе (УАПК) (ООО «БИОЧИП-ИМБ», Россия) для анализа биологических микрочипов с использованием специализированного программного обеспечения Image Ware® (ООО «БИОЧИП-ИМБ»). Гибридизационные картины приведены на Фиг. 2-22.
В соответствии с описанным выше алгоритмом интерпретации результатов, полученных на ДНК-чипе, устанавливали принадлежность анализируемой ДНК к видам микроорганизмов, являющихся возбудителями ИПНРФ (Т. pallidum, N. gonorrhoeae, С. trachomatis, М. genitalium, М. hominis, G. vaginalis, U. parvum, U. urealyticum, A. vaginae, E. faecalis, T. vaginalis, E. coli, M. mulieris, S. anginosus, F. nucleatum, S. epidermidis, B. fragilis), и определяли наличие генетических детерминат резистентности к антимикробным препаратам, включающим антибиотики пенициллинового ряда, цефалоспорины, тетрациклины, фторхинолоны, макролиды, нитроимидазолы, спектиномицин
Правильность идентификации генетических детерминант резистентности возбудителей ИПНРФ к антимикробным препаратам с использованием предлагаемого ДНК-чипа во всех случаях проверяли методом секвенирования соответствующих фрагментов генома возбудителя. Секвенирование проводили на автоматическом анализаторе 3130xL Applied Biosystems (США) с помощью набора «BigDye Terminator v3.1 Ready Reaction Cycle Sequencing Kit». Результаты, полученные с использованием ДНК-чипа, предлагаемого в данном изобретении, и референсного метода, совпали для всех анализируемых образцов.
Таким образом, ДНК-чип, представленный в данном изобретении, является инструментом для проведения мультиплексного анализа, который позволяет за короткое время точно определять генетические детерминанты резистентности возбудителей ИПНРФ к актуальным антибактериальным препаратам в клиническом материале. ДНК-чип выгодно отличается от существующих в настоящее время методов определения антибиотикорезистентности возможностью одновременного анализа 17 возбудителей ИПНРФ, широким спектром одновременно выявляемых детерминант резистентности, скоростью проведения анализа. Области применения ДНК-чипа включают медицину, в том числе дерматовенерологию, урологию, гинекологию, клиническую лабораторную диагностику, эпидемиологию.















































Изобретение относится к области биотехнологии, биохимии, молекулярной биологии, микробиологии и медицине. Предложен ДНК-чип и набор для идентификации генетических детерминант резистентности микроорганизмов - возбудителей инфекций, приводящих к нарушению репродуктивных функций человека: Treponema pallidum, Neisseria gonorrhoeae, Chlamydia trachomatis, Mycoplasma genitalium, Mycoplasma hominis, Gardnerella vaginalis, Ureaplasma parvum, Ureaplasma urealiticum, Atopobium vaginae, Enterococcus faecalis, Trichomonas vaginalis, Escherichia coli, Mobiluncus mulieris, Streptococcus anginosus, Fusobacterium nucleatum, Staphylococcus epidermidis, Bacteroides fragilis, к антимикробным препаратам, включающим антибиотики пенициллинового ряда, цефалоспорины, тетрациклины, фторхинолоны, макролиды, нитроимидазолы, спектиномицин. Изобретение может быть использовано в медицине для эффективной персонифицированной терапии заболеваний, клинической лабораторной диагностике, эпидемиологии. 2 н.п. ф-лы, 22 ил., 4 табл., 2 пр.
1. ДНК-чип для идентификации генетических детерминант резистентности микроорганизмов - возбудителей инфекций, приводящих к нарушению репродуктивных функций человека: Treponema pallidum, Neisseria gonorrhoeae, Chlamydia trachomatis, Mycoplasma genitalium, Mycoplasma hominis, Gardnerella vaginalis, Ureaplasma parvum, Ureaplasma urealiticum, Atopobium vaginae, Enterococcus faecalis, Trichomonas vaginalis, Escherichia coli, Mobiluncus mulieris, Streptococcus anginosus, Fusobacterium nucleatum, Staphylococcus epidermidis, Bacteroides fragilis, к антимикробным препаратам, включающим антибиотики пенициллинового ряда, цефалоспорины, тетрациклины, фторхинолоны, макролиды, нитроимидазолы, спектиномицин, характеризующийся тем, что он представляет собой матрицу из 168 ячеек, размещенных на подложке из стекла или пластика, причем матрица включает:
- 33 ячейки с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 1-33, соответствующими 16S рРНК Т. pallidum, N. gonorrhoeae, С.trachomatis, М. genitalium, М. hominis, G. vaginalis, U. parvum, U. urealiticum, A. vaginae, E. faecalis, T. vaginalis, E.coli, M. mulieris, S. anginosus, F. nucleatum, S. epidermidis, B. fragilis, для установления принадлежности анализируемой ДНК к перечисленным выше видам микроорганизмов, являющимся возбудителями инфекций, приводящих к нарушению репродуктивных функций человека;
- 9 ячеек с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 34-42, соответствующими последовательностям гена gyrA изолятов N. gonorrhoeae дикого типа либо последовательностям мутантных вариантов гена gyrA, приводящих к аминокислотным заменам S91F, S91T, D95N, D95G, встречающихся у изолятов N. gonorrhoeae, резистентных к фторхинолонам;
- 13 ячеек с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 43-55, соответствующими
последовательностям гена gyrA изолятов Е. coli дикого типа либо последовательностям мутантных вариантов гена gyrA, приводящих к аминокислотным заменам S83L, S83W, S83A, D87N, D87G, D87Y, встречающихся у изолятов Е. coli, резистентных к фторхинолонам;
- 3 ячейки с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 56-58, соответствующими последовательностям гена gyrA изолятов S. epidermidis дикого типа либо последовательностям мутантных вариантов гена gyrA, приводящих к аминокислотным заменам S83T, S83F, встречающихся у изолятов S. epidermidis, резистентных к фторхинолонам;
- 5 ячеек с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 59-63, соответствующими последовательностям гена gyrA изолятов F. nucleatum дикого типа либо последовательностям мутантных вариантов гена gyrA, приводящих к аминокислотным заменам S83L, S83T, G87R, встречающихся у изолятов F. nucleatum, резистентных к фторхинолонам;
- 4 ячейки с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 64-67, соответствующими последовательностям гена gyrA изолятов Е. faecalis дикого типа либо последовательностям мутантных вариантов гена gyrA, приводящих к аминокислотным заменам S83T, S83I, S83R, встречающихся у изолятов Е. faecalis, резистентных к фторхинолонам;
- 6 ячеек с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID 68-73, соответствующими последовательностям гена gyrA изолятов S. anginosus дикого типа либо последовательностям мутантных вариантов гена gyrA, приводящих к аминокислотным заменам S83T, S83F, встречающихся у изолятов S. anginosus, резистентных к фторхинолонам;
- 9 ячеек с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 74-82, соответствующими
последовательностям гена parC изолятов N. gonorrhoeae дикого типа либо последовательностям мутантных вариантов гена parC, приводящих к аминокислотным заменам S87N, S87R, E91Q, E91G, E91K, Е91А, встречающихся у изолятов N. gonorrhoeae, резистентных к фторхинолонам;
- 8 ячеек с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 83-90, соответствующими последовательностям гена parС изолятов М. genitalium дикого типа либо последовательностям мутантных вариантов гена parC, приводящих к аминокислотным заменам S83N, S83I, S83R, D87N, D87Y, D87V, встречающихся у изолятов М. genitalium, резистентных к фторхинолонам;
- 5 ячеек с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 91-95, соответствующими последовательностям гена parC изолятов М. hominis дикого типа либо последовательностям мутантных вариантов гена parC, приводящих к аминокислотным заменам S83I, S83N, E87K, встречающихся у изолятов М hominis, резистентных к фторхинолонам;
- 4 ячейки с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 96-99, соответствующими последовательностям гена parC изолятов U. urealiticum дикого типа либо последовательностям мутантных вариантов гена parC, приводящих к аминокислотным заменам D82N, S83L, встречающихся у изолятов U. urealiticum, резистентных к фторхинолонам;
- 4 ячейки с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 100-103, соответствующими последовательностям генов ntr4tv и ntr6tv изолятов Т. vaginalis дикого типа либо последовательностям их мутантных вариантов с нуклеотидными заменами C213G (Y71STOP) в гене ntr4tv, А238Т (K80STOP) в гене ntr6tv, встречающихся у изолятов Т. vaginalis, резистентных к нитроимидазолам;
- 6 ячеек с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 104-109, соответствующими
последовательностям генов nimB, nimC, nimD, nimE, nimF, nimG, наличие которых приводит к резистентности микроорганизма(ов) к нитроимидазолам;
- ячейку с иммобилизованным олигонуклеотидом с нуклеотидной последовательностью SEQ ID NO: 110, соответствующей последовательности плазмидных генов blaSHV-12 blaLEN-17, наличие которых приводит к резистентности микроорганизма(ов) к антибиотикам пенициллинового ряда и цефалоспоринам;
- ячейку с иммобилизованным олигонуклеотидом с нуклеотидной последовательностью SEQ ID NO: 111, соответствующей последовательности плазмидного гена blaSHV-97, наличие которого приводит к резистентности микроорганизма(ов) к антибиотикам пенициллинового ряда;
- 6 ячеек с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 112-117, соответствующими последовательностям плазмидных генов blaТEM и blaSHV, наличие которых приводит к резистентности микроорганизма(ов) к антибиотикам пенициллинового ряда, либо последовательностям мутантного варианта гена blаТЕМ с аминокислотными заменами М182Т и G238S, одновременное наличие которых приводит к резистентности микроорганизма(ов) к антибиотикам пенициллинового ряда и цефалоспоринам;
- 2 ячейки с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 118-119, соответствующими последовательностям гена penA изолятов N. gonorrhoeae дикого типа либо последовательностям мутантного варианта гена penA, приводящего к инсерции аспарагиновой кислоты (D) в положение 345, встречающегося у изолятов N. gonorrhoeae, резистентных к антибиотикам пенициллинового ряда;
- 4 ячейки с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 120-123, соответствующими последовательностям гена ponA изолятов N. gonorrhoeae дикого типа либо
последовательностям мутантного варианта гена ponA с аминокислотной заменой L421P, встречающейся у изолятов N. gonorrhoeae, резистентных к антибиотикам пенициллинового ряда;
- 3 ячейки с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 124-126, соответствующими последовательностям гена rpsJ изолятов N. gonorrhoeae дикого типа либо последовательностям мутантного варианта гена rpsJ с аминокислотной заменой V57M, V57L, встречающихся у изолятов N. gonorrhoeae, резистентных к тетрациклинам;
- 2 ячейки с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 127-128, соответствующими последовательностям генов tetM, наличие которых у Е. faecalis, E.coli, М. genitalium, М. hominis, N. gonorrhoeae, G. vaginalis приводит к резистентности к тетрациклинам;
- ячейку с иммобилизованным олигонуклеотидом с нуклеотидной последовательностью SEQ ID NO: 129, соответствующей последовательности гена tetO, наличие которого у изолятов Е. faecalis, S. epidermidis приводит к резистентности к тетрациклинам;
- 10 ячеек с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 130-139, соответствующими последовательностям 23S РНК изолятов U. urealiticum, U. hominis, S. anginosus, F. nucleatum, E. faecali, T. pallidum, E.coli, N. gonorrhoeae, C. trachomatis, M. genitalium, M. hominis, S. epidermidis, B. fragilis дикого типа либо последовательностям мутантных вариантов с заменами G2057A, A2058G, А2058С, A2059G, встречающихся у изолятов перечисленных возбудителей, резистентных к макролидам;
- 2 ячейки с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 140-141, соответствующими последовательности гена mefA, наличие которого приводит к резистентности S. anginosus, N. gonorrhoeae, S. epidermidis, В. fragilis к макролидам;
- 9 ячеек с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 142-150, соответствующими последовательностям 16S РНК изолятов N. gonorrhoeae дикого типа либо последовательностям мутантных вариантов с заменами С1192Т, G1064C, встречающихся у изолятов N. gonorrhoeae, резистентных к спектиномицину;
- 6 ячеек с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 148, 151-155, соответствующими последовательностям 16S РНК изолятов С.trachomatis, Е. coli, F. nucleatum, A. vaginae, Т. pallidum, U. urealiticum, U. hominis, S. anginosus, S. epidermidis, M. genitalium, M. hominis, M. mulieris, B. fragilis дикого типа либо последовательностям мутантных вариантов с заменами G1058C, G1064C, встречающихся у изолятов перечисленных возбудителей, резистентных к тетрациклину;
- 6 ячеек с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 156-161, соответствующими последовательностям гена помпы эффлюкса mtrR дикого типа либо последовательностям мутантных вариантов промоторной области гена mtrR, приводящих к делеции аденина А в положении -35 или инсерциям тимидина Т или ТТ в положении -10, встречающихся у изолятов возбудителей, резистентных к антибиотикам пенициллинового ряда, тетрациклинам, макролидам, цефалоспоринам;
- 2 ячейки с иммобилизованными олигонуклеотидами с нуклеотидными последовательностями SEQ ID NO: 162-163, соответствующими последовательности гена поринового белка porB изолятов N. gonorrhoeae дикого типа либо последовательности мутантного варианта гена porB, приводящей к аминокислотной замене G120К, встречающегося у изолятов N. gonorrhoeae, резистентных к антибиотикам пенициллинового ряда, цефалоспоринам, тетрациклинам.
2. Набор, состоящий из последовательностей олигонуклеотидных зондов с SEQ ID NO 1-163, используемый в ДНК-чипе по п. 1 для идентификации
генетических детерминант резистентности микроорганизмов - возбудителей инфекций, приводящих к нарушению репродуктивных функций человека: Treponema pallidum, Neisseria gonorrhoeae, Chlamydia trachomatis, Mycoplasma genitalium, Mycoplasma hominis, Gardnerella vaginalis, Ureaplasma parvum, Ureaplasma urealiticum, Atopobium vaginae, Enterococcus faecalis, Trichomonas vaginalis, Escherichia coli, Mobiluncus mulieris, Streptococcus anginosus, Fusobacterium nucleatum, Staphylococcus epidermidis, Bacteroides fragilis, к антимикробным препаратам, включающим антибиотики пенициллинового ряда, цефалоспорины, тетрациклины, фторхинолоны, макролиды, нитроимидазолы, спектиномицин.
| WO 2010062001 A1, 03.06.2010 | |||
| Универсальное двухствольное охотничье ружье | 1958 |
|
SU117431A1 |
| HENRIK BJORN NIELSEN et al | |||
| Design of oligonucleotides for microarrays and perspectives for design of multi-transcriptome arrays, Nucleic Acids Research, 2003, Vol | |||
| Способ очистки нефти и нефтяных продуктов и уничтожения их флюоресценции | 1921 |
|
SU31A1 |
| Насос | 1917 |
|
SU13A1 |
| BORIS SHASKOLSKIY et al., Drug Resistance Mechanismsin Bacteria Causing Sexually Transmitted Diseasesand Associatedwith Vaginosis, Frontiers in Microbiology, May 2016, Volume7, Article747, pp.1-14. | |||
Авторы
Даты
2019-04-16—Публикация
2016-11-25—Подача