Область техники, к которой относится изобретение
Изобретение относится к области молекулярной биологии, микробиологии, эпидемиологии и представляет собой способ идентификации однонуклеотидных полиморфизмов в многокопийном локусе генома методом петлевой полимеразной цепной реакции с использованием биологического микрочипа с иммобилизованными олигонуклеотидными зондами.
Уровень техники
Метод амплификации нуклеиновых кислот (НК) с помощью полимеразной цепной реакции (ПЦР) в настоящее время является важнейшим инструментом молекулярной биологии, и используются повсеместно во многих биологических и медицинских сферах (Green M.R. and Sambrook J. "Polymerase Chain Reaction"; Cold Spring Harb Protoc. 2019, v. 2019 (6), p. doi:10.1101/pdb.top095109). Метод основан на многократном избирательном копировании определенного участка НК, и при этом происходит копирование только того участка, который удовлетворяет заданным условиям.
В геноме различных организмов могут встречаться локусы, которые представлены в нескольких копиях, например гены рибосомной РНК, мобильные генетические элементы и т.д (Espejo R.T. and Plaza N. "Multiple Ribosomal RNA Operons in Bacteria; Their Concerted Evolution and Potential Consequences on the Rate of Evolution of Their 16S rRNA"; Front Microbiol. 2018, v. 9 p. 1232, doi:10.3389/fmicb.2018.01232). Следует отметить, что такие многокопийные локусы содержат однонуклеотидные полиморфизмы, и в одном геноме могут находиться одновременно как копии локуса дикого типа, так и копии с мутациями. Выявить однонуклеотидный полиморфизм в каждой отдельной копии с помощью классической ПЦР является непростой задачей, поскольку в этом случае все копии локуса будут удовлетворяет заданным условиям амплификации и выступать в качестве матрицы. Возникает потребность в проведении более специфичной амплификации, которая будет направлена на получение ПЦР-продуктов только с одной копии исследуемого локуса с последующим анализом последовательности локуса и идентификацией полиморфизма. Таким образом, необходимо провести параллельно несколько реакций амплификации для каждой отдельной копии исследуемого локуса, что может потребовать большего количества времени и реактивов (Marmesat Е., Soriano L. et al. "PCR Strategies for Complete Allele Calling in Multigene Families Using High-Throughput Sequencing Approaches"; PLoS One. 2016, v. 11 (6), p. e0157402, doi:10.1371/journal.pone.0157402).
Идентификация однонуклеотидного полиморфизма в многокопийном локусе генома может быть клинически важной задачей. Так, значимыми генетическими детерминантами устойчивости Neisseria gonorrhoeae к макролидам являются мутации в пептидилтрансферазной петле II и V домена 23S рРНК (Chisholm S.A., Dave J. et al. "High-level azithromycin resistance occurs in Neisseria gonorrhoeae as a result of a single point mutation in the 23S rRNA genes"; Antimicrob Agents Chemother. 2010, v. 54 (9), p. 3812-3816, doi:10.1128/AAC.00309-10). Замена C2611T приводит к появлению изолятов с умеренной резистентностью (значение минимальной подавляющей концентрации, МПК до 4 мг/л), в то время как мутации A2058G/A2059G ассоциированы с возникновением высокорезистентных штаммов (МПК > 256 мг/л) (Whiley D.M., Kundu R.L. et al. "Azithromycin-resistant Neisseria gonorrhoeae spreading amongst men who have sex with men (MSM) and heterosexuals in New South Wales, Australia, 2017"; J Antimicrob Chemother. 2018, v. 73 (5), p. 1242-1246, doi:10.1093/jac/dky017). Следует отметить, геном N. gonorrhoeae содержит многокопийный локус rrn (четыре копии), в состав которого входит ген 23 S рРНК, при этом уровень устойчивости определяется не только полиморфизмами в определенном положении гена 23 S рРНК, но еще и количеством мутантных копий данного локуса. В частности показано, что изоляты с тремя или со всеми четырьмя мутантными копиями являются высокорезистентными (МПК > 256 мг/л и до 4096 мкг/мл), а изоляты только с одной копией мутантного гена 23S рРНК приводят к незначительному повышению МПК с 0,25 до 1 мг/л, что ниже или на уровне порогового значения МПК=1 мг/л, дифференцирующего чувствительные и устойчивые к азитромицину формы возбудителя гонококковой инфекции (Zhou Q., Liu J. et al. "The Accuracy of Molecular Detection Targeting the Mutation C2611T for Detecting Moderate-Level Azithromycin Resistance in Neisseria gonorrhoeae: A Systematic Review and Meta-Analysis"; Antibiotics (Basel). 2021, v. 10 (9), p. doi: 10.3390/antibiotics 10091027).
Для выявления однонуклеотидных полиморфизмов в многокопийном локусе генома предложены методы амплификации нуклеиновых кислот с различными способами детекции. Описан метод «вложенной ПЦР» в котором в первой стадии амплификации используются праймеры, специфичные к фланкирующей последовательности для каждой из копий, затем в новой пробирке проводится второй этап амплификации, после чего проводят секвенирование или рестрикционный анализ для определения нуклеотидной последовательности каждой копии (Su С., Shwab Е.K. et al. "Moving towards an integrated approach to molecular detection and identification of Toxoplasma gondii"; Parasitology. 2010, v. 137 (1), p. 1-11, doi: 10.1017/S0031182009991065).
Описана система идентификации однонуклеотидных полиморфизмов с помощью ПЦР в реальном времени (Kalendar R., Shustov A.V. et al. "Designing Allele-Specific Competitive-Extension PCR-Based Assays for High-Throughput Genotyping and Gene Characterization"; Front Mol Biosci. 2022, v. 9 p. 773956, doi: 10.3389/fmolb.2022.773956). Данная система требует предварительной реакции амплификации, где используется один обратный праймер, комплементарный всем копиям исследуемого локуса, и набор прямых праймеров, которые состоят из нескольких частей и комплементарны только одной копии исследуемого локуса. Далее проводится второй этап ПЦР в реальном времени, где в качестве матрицы используются полученные на предварительной стадии ПЦР-продукты, и в зависимости от канала флуоресценции устанавливается наличие или отсутствие исследуемого полиморфизма в каждой копии. Также описан способ определения однонуклеотидных полиморфизмов методом ПЦР с флуоресцентной детекцией накопления ДНК во время реакции (патент РФ №2005135742, дата приоритета 18.11.2005 г., опубликовано 27.05.2007 г.). Недостатком таких систем является необходимость проведения реакции амплификации каждой копии локуса в отдельном реакционном объеме (отдельной пробирке). Другим ограничением является тот факт, что количество исследуемых копий локуса лимитировано числом флуоресцентных каналов амплификатора (до пяти).
Выявление однонуклеотидных полиморфизмов в многокопийном локусе может быть осуществлено с помощью технологии полногеномного секвенирования, которая не основана на реакции амплификации и позволяет получать длинные прочтения, например, технологии Oxford Nanopore или Pacific Biosciences (Moolhuijzen P., See P.T. et al. "PacBio genome sequencing reveals new insights into the genomic organisation of the multi-copy ToxB gene of the wheat fungal pathogen Pyrenophora tritici-repentis"; BMC Genomics. 2020, v. 21 (1), p. 645, doi: 10.1186/s12864-020-07029-4, Nowak A., Murik O. et al. "Detection of single nucleotide and copy number variants in the Fabry disease-associated GLA gene using nanopore sequencing"; Sci Rep. 2021, v. 11 (1), p. 22372, doi:10.1038/s41598-021-01749-7). При получении длинных прочтений можно установить принадлежность каждого полиморфизма к конкретной копии, а также определить число копий и их положение в геноме (Istace В., Friedrich A. et al. "de novo assembly and population genomic survey of natural yeast isolates with the Oxford Nanopore MinION sequencer"; Gigascience. 2017, v. 6 (2), p. 1-13, doi:10.1093/gigascience/giw018). Недостатком данного метода является сложность в подготовке образцов для секвенирования, дороговизна эксперимента, что пока не позволяет рассматривать данный способ для рутинного применения.
Из анализа уровня техники можно сделать вывод, что в настоящее время существует необходимость разработки способа идентификации однонуклеотидных полиморфизмов в многокопийном локусе генома, который выгодно отличался бы от известных способов возможностью одновременной идентификации полиморфизмов сразу во всех копиях исследуемого локуса, а также обладал достаточной простотой и скоростью проведения анализа.
Раскрытие изобретения
В настоящем изобретении предложен способ идентификации однонуклеотидных полиморфизмов, который выгодно отличается от известных из уровня техники изобретений тем, что позволяет проводить этапы амплификации в едином реакционном объеме, что избавляет от необходимости проведения нескольких последовательных стадий ПЦР с переносом продуктов реакции из одного объема в следующий, а также позволяет идентифицировать однонуклеотидный полиморфизм в каждой отдельной копии исследуемого локуса, существенно сокращая время проведения эксперимента. Способ не требует дорогостоящего оборудования и высококвалифицированного персонала.
Предлагаемый способ идентификации однонуклеотидных полиморфизмов основан на проведении петлевой ПЦР с получением одноцепочечных флуоресцентно-меченных ПЦР-фрагментов, с последующей гибридизацией полученных фрагментов на биологическом микрочипе (биочипе, микрочипе), регистрацией гибридизационной картины (флуоресцентного изображения) и анализом флуоресцентных сигналов элементов биочипа. Интерпретация результатов осуществляется в зависимости от того, в каком элементе биочипа зарегистрированы совершенные гибридизационные комплексы, образованные иммобилизованным олигонуклеотидным зондом и ПЦР-фрагментом, включающим одну из копий исследуемого локуса. Способ включает следующие стадии:
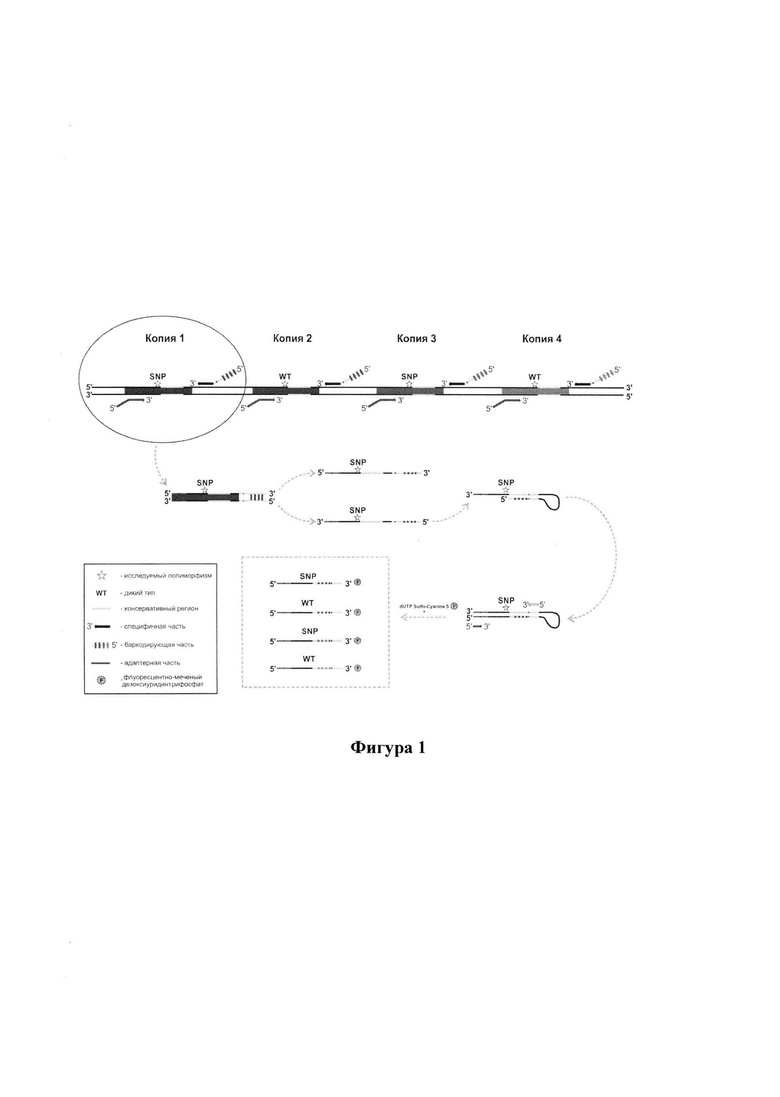
а) этап амплификации, в котором в качестве матрицы используют геномную ДНК, включающую многокопийный локус с исследуемым полиморфизмом и консервативным регионом, а в качестве праймеров используют один прямой праймер, последовательность которого состоит из двух частей - 3'-комплементарной последовательности фрагмента исследуемого локуса и 5'-«адаптерной», последовательность которой некомплементарна последовательности исследуемого локуса, и набор обратных праймеров, последовательности которых состоят из двух частей - 3'-специфичной, комплементарной последовательности фрагмента, фланкирующего отдельную копию исследуемого локуса, и 5'-«баркодирующей», комплементарной последовательности консервативного региона исследуемого локуса и содержащей уникальный для каждой копии исследуемого локуса набор однонуклеотидных замен (баркодов); тем самым в ходе этапа амплификации образуются ПЦР-продукты, ограниченные «адаптерной» последовательностью прямого праймера и «баркодирующими» последовательностями обратных праймеров;
б) этап амплификации, в котором в одной из цепей ПЦР-продукта, полученного на стадии (а), 5'-«баркодирующая» последовательность гибридизуется с комплементарной ей последовательностью консервативного региона исследуемого локуса, что обеспечивает образование и накопление продуктов с вторичной структурой типа «петля» с использованием праймеров стадии (а);
в) этап амплификации, в котором в качестве матрицы используют ПЦР-продукты с вторичной структурой типа «петля», полученные на стадии (б), в качестве прямого праймера используют праймер, последовательность которого комплементарна «адаптерной» части прямого праймера, используемого на стадии (а), в качестве обратного праймера используют праймер, последовательность которого комплементарна не содержащему «баркодов» фрагменту «баркодирующей» части обратного праймера, используемого на стадии (а); при этом концентрации прямого и обратного праймеров в реакционной смеси могут различаться в 10 и более раз, а в реакционную смесь добавляют флуоресцентно-меченый дезоксиуридинтрифосфат с целью получения преимущественно одноцепочечных флуоресцентно-меченных ПЦР-продуктов, включающих последовательность локуса с однонуклеотидным полиморфизмом и последовательность «баркодирующей» части, соответствующую отдельной копии исследуемого локуса;
г) обеспечение биологического микрочипа для идентификации однонуклеотидного полиморфизма в исследуемом локусе, представляющего собой матрицу элементов, в каждом из которых иммобилизован уникальный олигонуклеотидный зонд, имеющий последовательность, комплементарную последовательности одноцепочечного фрагмента, полученного на стадии (в), и выбранную из группы, включающей: I) последовательности локуса дикого типа и фрагмента «баркодирующей» части, соответствующей отдельной копии исследуемого локуса; II) последовательности локуса с заменой, характеризующей однонуклеотидный полиморфизм, и фрагмента соответствующей отдельной копии исследуемого локуса;
д) гибридизацию амплифицированных флуоресцентно-меченных ПЦР-продуктов, полученных на стадии (в), на биологическом микрочипе, полученном на стадии (г), с образованием совершенных и несовершенных дуплексов с иммобилизованными олигонуклеотидными зондами;
е) регистрацию результатов гибридизации на биологическом микрочипе, проведенной на стадии (д) с использованием портативного анализатора флуоресценции и программного обеспечения, что позволяет использовать программную обработку интенсивностей сигналов с последующей интерпретацией результатов.
В одном из воплощений способ характеризуется тем, что разница в температурах отжига праймеров, используемых на стадиях (а) и (в), составляет не менее 10°С, что позволяет проводить этапы амплификации (а)-(в) в едином реакционном объеме последовательно при разных температурах отжига.
В другом воплощении способ характеризуется тем, что на стадии (е) совершенные дуплексы регистрируют в элементах микрочипа в зависимости от последовательности «баркодирующей» части, что позволяет идентифицировать однонуклеотидный полиморфизм в каждой отдельной копии исследуемого локуса.
Краткое описание фигур и таблиц
Фигура 1. Схема петлевой полимеразной цепной реакции с целью одновременной амплификации четырех копий локуса, в ходе которой образуются одноцепочечные флуоресцентно-меченные ПЦР-продукты. Этапы амплификации обозначены серыми пунктирными линиями. Копии локуса (1-4) обозначены разными цветами, и при этом вторая и четвертая копия не содержат полиморфизмов в исследуемом положении, т.е являются копиями дикого типа (WT), а первая и третья копии содержат исследуемый полиморфизм (SNP).
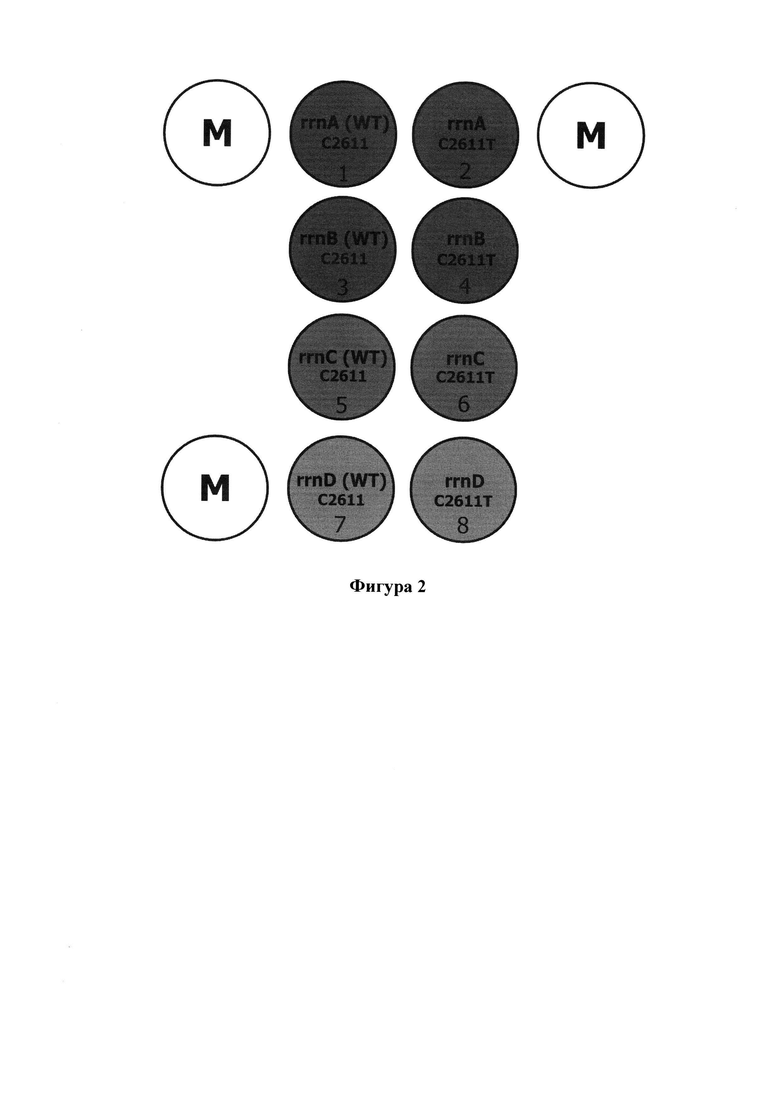
Фигура 2. Схема размещения элементов биологического микрочипа (биочипа) для идентификации однонуклеотидного полиморфизма в положении 2611 в многокопийном локусе 23S рРНК генома N. gonorrhoeae.
Фигура 3. Гибридизационная картина (флуоресцентное изображение) биологического микрочипа (биочипа) при анализе ДНК изолята N. gonorrhoeae (МПКазм=0,12 мг/л), не содержащего полиморфизма в положении 2611 во всех четырех копиях гена 23 S рРНК. Фигура 4. Гибридизационная картина (флуоресцентное изображение) биологического микрочипа (биочипа) при анализе ДНК изолята N. gonorrhoeae (МПКазм=2 мг/л), содержащего полиморфизм С2611Т в первой и третьей копии гена 23S рРНК. Фигура 5. Гибридизационная картина (флуоресцентное изображение) биологического микрочипа (биочипа) при анализе ДНК изолята N. gonorrhoeae (МПКазм=4 мг/л), содержащего полиморфизм С2611Т во всех четырех копиях гена 23 S рРНК. Таблица 1. Перечень праймеров, используемых для идентификации однонуклеотидных полиморфизмов в положении 2611 гена 23S рРНК N. gonorrhoeae методом петлевой ПЦР. Прямой праймер SEQ ID NO: 1 для этапа амплификации (а) состоит из двух частей - 3'-комплементарной последовательности фрагмента гена 23 S рРНК (обычным шрифтом), и 5'-«адаптерной», последовательность которой некомплементарна последовательности исследуемого локуса 23S рРНК (жирным шрифтом). Обратные праймеры (SEQ ID NO: 2-5) для этапа амплификации (а) состоят из двух частей - 3'-специфичной, комплементарной последовательности фрагмента, фланкирующего отдельную копию исследуемого гена 23S рРНК (курсивом), и 5'-«баркодирующей» (обычным и жирным шрифтом), комплементарной последовательности консервативного региона исследуемого локуса и содержащей уникальный для каждой копии исследуемого локуса набор однонуклеотидных замен (баркодов) (подчеркнуты). Прямой праймер SEQ ID NO: 6 для этапа амплификации (б) имеет последовательность, комплементарную «адаптерной» части прямого праймера SEQ ID NO: 1 (жирным шрифтом). Обратный праймер SEQ ID NO: 7 для этапа амплификации (б) имеет последовательность, комплементарную не содержащему «баркодов» фрагменту «баркодирующей» части обратных праймеров (SEQ ID NO: 2-5) (жирным шрифтом), используемых на стадии (а).
Таблица 2. Перечень олигонуклеотидных зондов, иммобилизованных в элементах биологического микрочипа (биочипа), используемого для идентификации однонуклеотидного полиморфизма в положении 2611 гена 23 S рРНК N. gonorrhoeae (SEQ ID NO: 8-15). Для каждой копии гена 23S рРНК N. gonorrhoeae создана пара зондов, включающая зонд, соответствующий гену дикого типа (WT), и зонд с заменой, характеризующий однонуклеотидный полиморфизм - замену цитозина (С) на тимин (Т) в положении 2611 гена 23 S рРНК. Каждый зонд имеет составную последовательность, 3'-часть которой (выделена жирным шрифтом) комплементарна последовательности гена 23 S рРНК N. gonorrhoeae дикого типа, либо с заменой С2611Т, а 5'-часть - комплементарна фрагменту с однонуклеотидными заменами (баркодами), соответствующими отдельной копии исследуемого локуса (баркоды подчеркнуты).
Осуществление изобретения
Целью изобретения являлось создание способа идентификации однонуклеотидных полиморфизмов в многокопийном локусе генома методом петлевой полимеразной цепной реакции с последующей гибридизацией на биологическом микрочипе. Предлагаемый способ позволяет проводить этапы амплификации в едином реакционном объеме, что позволяет избежать нескольких последовательных стадий ПЦР с переносом продуктов реакции из одного объема в следующий и идентифицировать однонуклеотидный полиморфизм в каждой отдельной копии исследуемого локуса, существенно сокращая время проведения эксперимента.
Заявленное изобретение включает петлевую ПЦР с целью амплификации всех копий локуса в едином реакционном объеме, схема которой представлена на Фигуре 1, включающую:
а) этап амплификации, в котором в качестве матрицы используют геномную ДНК, включающую многокопийный локус с исследуемым полиморфизмом и консервативным регионом, а в качестве праймеров используют один прямой праймер, последовательность которого состоит из двух частей - 3'-комплементарной последовательности фрагмента исследуемого локуса и 5'-«адаптерной», последовательность которой некомплементарна последовательности исследуемого локуса, и набор обратных праймеров, последовательности которых состоят из двух частей - 3'-специфичной, комплементарной последовательности фрагмента, фланкирующего отдельную копию исследуемого локуса, и 5'-«баркодирующей», комплементарной последовательности консервативного региона исследуемого локуса и содержащей уникальный для каждой копии исследуемого локуса набор однонуклеотидных замен (баркодов); тем самым в ходе этапа амплификации образуются ПЦР-продукты, ограниченные «адаптерной» последовательностью прямого праймера и «баркодирующими» последовательностями обратных праймеров;
б) этап амплификации, в котором в одной из цепей ПЦР-продукта, полученного на стадии (а), 5'-«баркодирующая» последовательность гибридизуется с комплементарной ей последовательностью консервативного региона исследуемого локуса, что обеспечивает образование и накопление продуктов с вторичной структурой типа «петля» с использованием праймеров стадии (а);
в) этап амплификации, в котором в качестве матрицы используют ПЦР-продукты с вторичной структурой типа «петля», полученные на стадии (б), в качестве прямого праймера используют праймер, последовательность которого комплементарна «адаптерной» части прямого праймера, используемого на стадии (а), в качестве обратного праймера используют праймер, последовательность которого комплементарна не содержащему «баркодов» фрагменту «баркодирующей» части обратного праймера, используемого на стадии (а); при этом концентрации прямого и обратного праймеров в реакционной смеси могут различаться в 10 и более раз, а в реакционную смесь добавляют флуоресцентно-меченый дезоксиуридинтрифосфат с целью получения преимущественно одноцепочечных флуоресцентно-меченных ПЦР-продуктов, включающих последовательность локуса с однонуклеотидным полиморфизмом и последовательность «баркодирующей» части, соответствующую отдельной копии исследуемого локуса;
Заявленное изобретение также включает обеспечение биологического микрочипа (биочипа) для идентификации однонуклеотидного полиморфизма в исследуемом локусе, представляющего собой матрицу элементов, в каждом из которых иммобилизован уникальный олигонуклеотидный зонд, имеющий последовательность, комплементарную последовательности одноцепочечного фрагмента, полученного на стадии (в), и выбранную из группы, включающей: а) последовательности локуса дикого типа и фрагмента «баркодирующей» части, соответствующей отдельной копии исследуемого локуса; б) последовательности локуса с заменой, характеризующей однонуклеотидный полиморфизм, и фрагмента соответствующей отдельной копии исследуемого локуса.
Матрица элементов размещается на подложке из стекла или пластика, которые могут быть сформированы разными способами. Растворы олигонуклеотидных зондов могут быть нанесены непосредственно на подложку с получением так называемого «планарного» биочипа, как описано в книге (Matson R.S. Microarray Methods and Protocols. 2009. CRC Press. 216 pages, ISBN 9781420046656).
Биочип также может представлять собой матрицу, состоящую из гидрогелевых элементов полусферической формы («трехмерный биочип»), содержащих ковалентно иммобилизованные олигонуклеотиды, изготовленный методом сополимеризационной иммобилизации, разработанной и запатентованный Институтом молекулярной биологии им. В.А. Энгельгардта Российской академии наук (ИМБ РАН) (Gryadunov D.A., Shaskolskiy B.L. et al. "The EIMB Hydrogel Microarray Technology: Thirty Years Later"; Acta Naturae. 2018, v. 10 (4), p. 4-18. doi: 10.32607/20758251-2018-10-4-4-18); (Shaskolskiy В., Kandinov I. et al. "Hydrogel Droplet Microarray for Genotyping Antimicrobial Resistance Determinants in Neisseria gonorrhoeae Isolates"; Polymers (Basel). 2021, v. 13 (22), 3889. doi:10.3390/polym13223889). При изготовлении гидрогелевого биочипа предварительно формируют композиции, содержащие гелеобразующие мономеры и олигонуклеотидные зонды, подлежащие иммобилизации. Композиции наносят на подложки из стекла или пластика в виде массива микрокапель с помощью автоматического микродозатора (робота). Под действием УФ-излучения происходит полимеризация геля с одновременной ковалентной иммобилизацией зондов в элементах (каплях объемом до 0,1 нл).
Заявленное изобретение также включает гибридизацию флуоресцентно-меченных продуктов на биочипе, с образованием совершенных и несовершенных дуплексов с иммобилизованными олигонуклеотидными зондами. Гибридизацию проводят в растворе, содержащем буферный компонент для поддержания рН, соль для создания ионной силы и хаотропный (дестабилизирующий водородные связи) агент, при температуре, зависящей от температуры плавления иммобилизованных на микрочипе олигонуклеотидов, например, как описано в работе (Mikhailovich V.M., Lapa S.A. et al. "Detection of rifampicin-resistant Mycobacterium tuberculosis strains by hybridization and polymerase chain reaction on a specialized ТВ-microchip"; Bull Exp Biol Med. 2001, v. 131 (1), p. 94-98, doi: 10.1023/a: 1017555318388).
На следующем этапе проводят регистрацию флуоресцентных сигналов элементов биочипа, в которых произошло формирование гибридизационных комплексов между иммобилизованными олигонуклеотидными зондами и флуоресцентно-меченными ПЦР-продуктами, включающими фрагмент локуса с однонуклеотидным полиморфизмом, и «баркодирующую» часть, соответствующую отдельной копии исследуемого локуса. Регистрацию выполняют с использованием анализатора флуоресценции биочипов (Lysov Yu.P., Barsky V.E. et al. 'Microarray analyzer based on wide field fluorescent microscopy with laser illumination and a device for speckle suppression'; Biomed. Opt. Express. 2017. v. 8 (11), p. 4798-4810, doi: 10.1364/BOE.8.004798).
Интерпретацию результатов гибридизации выполняют посредством сравнения интенсивностей флуоресцентного сигнала элементов, в которых образовались совершенные и несовершенные гибридизационные комплексы. Сравнение флуоресцентных сигналов проводят внутри каждой отдельной группы зондов. Каждая группа элементов соответствует отдельной копии исследуемого локуса и включает зонд, последовательность которого выбрана из последовательности дикого типа, и зонд, последовательность которого выбрана из последовательности локуса с заменой, характеризующей однонуклеотидный полиморфизм. Максимальный сигнал элемента свидетельствует о наличии или отсутствии однонуклеотидного полиморфизма в каждой отдельной копии анализируемого локуса.
Описанный алгоритм интерпретации результатов гибридизации может быть реализован в программном обеспечении, позволяющем проводить автоматическую обработку результатов при регистрации флуоресцентного изображения биочипа, определения интенсивности флуоресцентного сигнала в каждом элементе, сравнения сигналов внутри групп и выдачи отчета о наличии или отсутствия однонуклеотидного полиморфизма в каждой отдельной копии анализируемого локуса.
Далее изобретение будет проиллюстрировано примером, который предназначен для обеспечения лучшего понимания сущности заявленного изобретения, но не должен рассматриваться как ограничивающий данное изобретение.
Пример. Идентификация однонуклеотидного полиморфизма С2611Т в многокопийном (до четырех копий) опероне rrn N. gonorrhoeae методом петлевой полимеразной цепной реакции с последующей гибридизацией на биологическом микрочипе. Процедура включала следующие этапы:
I. Получение геномной ДНК возбудителя гонореи N. gonorrhoeae
Использовали клинические изоляты из коллекции Государственного научного центра дерматовенерологии и косметологии Минздрава РФ (Kubanov A., Solomka V. et al. "Summary and Trends of the Russian Gonococcal Antimicrobial Surveillance Programme, 2005 to 2016"; J Clin Microbiol. 2019, v. 57 (6), e02024-18. doi:10.1128/JCM.02024-18). Всего было использовано три клинических изолята: №1 с МПКазм=0,12 мг/л; №2 с МПКАзм=2 мг/л; №3 с МПКАзм=4 мг/л.
Изоляты высевают на шоколадный агар GC (Difco, США) с добавлением ростовых добавок IsoVitaleX Enrichmen (BD, США). Чашку петри инкубируют ночь при температуре 37 С°, 5% СО2. С помощью культуральной петли собирают выросшие колонии и суспендируют их в 100 мкл PBS буфера. Выделение ДНК проводят с использованием экспресс-наборов для выделения ДНК НПФ «Литех» (Россия). Конечные концентрации ДНК измеряют с помощью спектрофотометра NanoDrop 2000 и флуориметра Qubit 4 (Thermo Scientific, USA), концентрации находились в пределах 20-100 нг/мкл. Геномная ДНК включала локус rrn, в состав которого входит ген 23S рРНК, представленный в четырех копиях, включая исследуемый полиморфизм С2611Т и примыкающий к нему консервативный регион.
II. Конструирование праймеров для ПЦР
Конструирование праймеров проводят в программе Bioedit (Ibis biosciences, США). В качестве референсных геномов используют 14 штаммов всемирной организации здравоохранения (ВОЗ) (Unemo М., Golparian D. et al. "The novel 2016 WHO Neisseria gonorrhoeae reference strains for global quality assurance of laboratory investigations: phenotypic, genetic and reference genome characterization"; J Antimicrob Chemother. 2016, v. 71 (11), p. 3096-3108, doi:10.1093/jac/dkw288). Устанавливают положение каждой отдельной копии гена 23 S рРНК в геноме, и определяют последовательности, фланкирующие отдельную копию. Позиции для праймеров выбирают с учетом GC состава последовательности и гомополимерных повторов. Температуры плавления Tm олигонуклеотидов рассчитывают с использованием программного обеспечения OligoAnalyzer Tool (Integrated DNA Technologies, https://www.idtdna.com/pages./tools/oligoanalyzer), и, варьируя их длину, добиваются того, чтобы разброс Tm составлял не более 2-3 С°. Избегают олигонуклеотидов, которые способны формировать вторичные структуры типа шпильки с высокими Tm (расчетная свободная энергия образования структуры ΔG ≤ - 6 ккал/моль). Праймеры для амплификации представлены в Таблице 1.
Для этапа амплификации (а) конструируют один прямой праймер (SEQ ID NO: 1 в Таблице 1), последовательность которого состоит из двух частей - 3'-комплементарной последовательности фрагмента гена 23 S рРНК (обычным шрифтом), и 5'-«адаптерной», последовательность которой некомплементарна последовательности исследуемого локуса 23S рРНК (жирным шрифтом). Также конструируют набор обратных праймеров (SEQ ID NO: 2-5 Таблице 1), последовательности которых состоят из двух частей - 3'-специфичной, комплементарной последовательности фрагмента, фланкирующего отдельную копию исследуемого гена 23 S рРНК (курсивом), и 5'-«баркодирующей» (обычным и жирным шрифтом), комплементарной последовательности консервативного региона исследуемого локуса и содержащей уникальный для каждой копии исследуемого локуса набор однонуклеотидных замен (баркодов) (подчеркнуты). Например, для первой копии гена 23 S рРНК баркодами являются: цитозин на пятом месте (5'-3') нуклеотидной последовательности баркодирующей части праймера, гуанин на седьмом месте, и цитозин на десятом месте (SEQ ID NO: 2 в Таблице 1).
Для этапа амплификации (б) конструируют прямой праймер (SEQ ID NO: 6 в Таблице 1), последовательность которого комплементарна «адаптерной» части прямого праймера (SEQ ID NO: 1 в Таблице 1) (жирным шрифтом), и обратный праймер (SEQ ID NO: 7 в Таблице 1), последовательность которого комплементарна не содержащему «баркодов» фрагменту «баркодирующей» части обратных праймеров (жирным шрифтом), используемых на стадии (a) (SEQ ID NO: 2-5 в Таблице 1).
III. Синтез праймеров
Праймеры получают путем стандартного фосфорамидитного синтеза со шкалой от 0,2 до 1 μМ на автоматическом синтезаторе MerMade 48Х (Biosearch Technologies, США). Очистку олигонуклеотидных праймеров проводят путем обращенно-фазовой ВЭЖХ на колонке Supelco LC-18 (5 мкм, 4,6 × 250 мм, Sigma, США) с элюцией или 0,05М ацетатом триэтиламмония (ТЕАА), рН 7,0, или 0,05М ТЕАА в градиенте концентраций CH3CN от 0 до 50%.
IV. Петлевая амплификация всех копий локуса rrn в едином реакционном объеме
В качестве матрицы используют геномную ДНК N. gonorrhoeae, полученную на этапе (I). Пробирка №1 содержала 3 мкл образца ДНК изолята с МПКазм=0,12 мг/л; пробирка №2 - 3 мкл образца ДНК изолята с МПКАзм=2 мг/л, пробирка №3 - 3 мкл образца ДНК изолята с МПКАзм=4 мг/л.
Готовят реакционную смесь, внося в каждую из пробирок следующие реагенты:
- 10х ПЦР-буфер для HotStarTaq Tersus ДНК-полимеразы (ЗАО «ЕВРОГЕН», Россия) - 3,0 мкл;
- дНТФ (водный раствор дезоксинуклеозидтрифосфатов, 2 мМ каждого) - 3,0 мкл;
- флуоресцентный субстрат - конъюгат дезоксиуридинтрифосфата и красителя Су5, 1 мМ - 0,2 мкл (ООО «ЛЮМИПРОБ РУС»);
- смесь праймеров для ПЦР: SEQ ID NO: 1-5 по 50 пмоль/мкл, праймер SEQ ID NO: 6 - 1 пмоль/мкл, праймер SEQ ID NO: 7 - 50 пмоль/мкл, всего - 3,0 мкл;
- HotStarTaq Tersus ДНК-полимераза (ЗАО «ЕВРОГЕН», Россия) - 1,0 мкл.
- вода деионизованная - 21,3 мкл;
ПЦР проводят на термоциклере S1000 (Bio-Rad, США) со следующим профилем:

По окончании ПЦР получают ПЦР-продукты, различающиеся последовательностями, зависящими от состава геномной ДНК анализируемых изолятов N.gonorrhoeae. Полученные растворы используют на этапе (VII) гибридизации на биологических микрочипах.
V. Конструирование и синтез зондов для иммобилизации на биочипе
Конструирование олигонуклеотидных зондов проводят в программе Bioedit (Ibis biosciences, США). Создают множественное выравнивание 14 референсных геномов N. gonorrhoeae, как описано в п. II. Определяют положение каждого из четырех копий гена 23S рРНК, включающего последовательность с полиморфизмом С2611Т. Список олигонуклеотидных зондов SEQ ID NO: 8-15 приведен в Таблице 2. Каждый зонд обладал составной последовательностью, 3'-часть которой (выделена жирным шрифтом) комплементарна последовательности гена 23 S рРНК N. gonorrhoeae дикого типа, либо с заменой, соответствующей полиморфизму в положении 2611, а 5'-часть - комплементарна фрагменту с однонуклеотидными заменами (баркодами), соответствующими отдельной копии исследуемого локуса (баркоды подчеркнуты). Для каждой копии гена 23 S рРНК N. gonorrhoeae конструируют пару зондов, включая зонд, соответствующий гену дикого типа (WT), и зонд с заменой С2611Т в гене 23S рРНК.
Используя программное обеспечение «Oligo 7» (Molecular Biology Insights Inc., США), рассчитывают температуры плавления олигонуклеотидов и, варьируя их длину, добиваются того, чтобы разброс температур плавления дискриминирующих олигонуклеотидных зондов внутри одной группы составлял не более 2-3°С. Избегают таких олигонуклеотидов, которые способны формировать вторичные структуры типа шпильки с высокими температурами плавления. Положение определяемых вариабельных нуклеотидов (С2611/С2611Т) выбирают по возможности не далее 1-4 нуклеотида от середины олигонуклеотидного зонда.
Олигонуклеотидные зонды получают путем стандартного фосфорамидитного синтеза со шкалой от 0,2 до 1 μМ на автоматическом синтезаторе MerMade 48Х (Biosearch Technologies, США). Модификацию аминогруппой 5'-конца выполняют с помощью 5'-Amino-Modifier С6 и Unilink™ Amino-Modifier (Glen Research, США). Очистку олигонуклеотидных зондов проводят путем обращенно-фазовой ВЭЖХ на колонке Supelco LC-18 (5 мкм, 4,6 х 250 мм, Sigma, США) с элюцией или 0,05М ацетатом триэтиламмония (ТЕАА), рН 7,0, или 0,05М ТЕАА в градиенте концентраций CH3CN от 0 до 50%.
VI. Изготовление гидрогелевых биочипов
Биочипы, содержащие олигонуклеотидные зонды, полученные, как описано в п.V, и иммобилизованные в гидрогеле, изготавливают в соответствии с описанными ранее методами (Shaskolskiy В., Kandinov I. et al. "Hydrogel Droplet Microarray for Genotyping Antimicrobial Resistance Determinants in Neisseria gonorrhoeae Isolates"; Polymers (Basel). 2021, v. 13 (22), 3889. doi:10.3390/polyml3223889). Композиции для нанесения включали 4,75% метакриламид, 0,25% N,N'-метиленбисакриламид, 50% глицерин, N,N,N',N'-5% тетраметилендиамин (TEMED) и олигонуклеотидные зонды в концентрации 2000 пмоль/мкл. Подготовленные в пробирках композиции переносят в 384-луночный планшет, и наносят на полимерные пластины (ООО «РИ-СК», Россия) в виде массива капель-полусфер объемом ~0,1 нл с помощью автоматического микродозатора - установки для переноса гидрогелей ИМБ РАН. Диаметр полусферических гелевых ячеек составлял 100 мкм, расстояние между центрами ячеек - также 100 мкм. Полимеризацию гелевых элементов проводят под действием УФ с максимальной длиной волны 350 нм и интенсивностью 0,06 μW/см2 в течение 40 мин при 20°С. После полимеризации пластины промывают 0,1 М буфером PBS, содержащим 0,1% Tween 20, затем деионизированной водой и высушивают при комнатной температуре. Разрезание полимеризационных пластин на индивидуальные подложки микрочипов выполняют с использованием лазера, фиксированного на двухкоординатном позиционере, после чего подложки вкладывают в пластиковые держатели формата предметного стекла и проводят технологический контроль качества. Проверку каждого микрочипа выполняют с использованием портативного флуоресцентного микроскопа, оснащенного CCD-камерой и программным обеспечением, распознающим элементы биочипа и обсчитывающим основные параметры (диаметр, объем, отклонения от заданных геометрических параметров матрицы) с последующей статистической обработкой и выдачей информации о пригодности микрочипа для дальнейшего использования. В случае положительного заключения проводят окончательную сборку микрочипа посредством монтажа составной гибридизационной камеры объемом 30 мкл (https://www.mdpi.com/polymers/polymers-13-03889/article_deploy/html/images/polymers-13-03889-ag.png) и используют готовый биочип для гибридизационного анализа. Схема размещения элементов биочипа для идентификации однонуклеотидного полиморфизма С2611Т в многокопийном локусе 23S рРНК, представлена на Фигуре 2.
VII. Гибридизация на биочипах
В отдельную пробирку помещают 10 мкл буфера для гибридизации (0,3 М HEPES, рН 7,5, 3,0 М гуанидинтиоцианата, 30 мМ ЭДТА) и 20 мкл реакционной смеси из пробирок №1-3, полученных, как описано в п. IV. Смеси объемом 30 мкл вносят в гибридизационные камеры биочипов, изготовленных, как описано в п. VI. Биочипы помещают в термостат и проводят гибридизацию при 37°С в течение 6-12 час. По окончании гибридизации биочипы трижды промывают дистиллированной водой и высушивают в потоке воздуха до полного исчезновения капель на поверхности подложки.
VIII. Регистрация и интерпретация результатов гибридизации
Для регистрации гибридизационных картин используют Универсальный аппаратно-программный комплекс для анализа биологических микрочипов (УАПК) (ООО «БИОЧИП-ИМБ», Россия), оснащенный специализированным программным обеспечением «ImageWare» (ООО «БИОЧИП-ИМБ»).
Интерпретацию результатов гибридизации выполняют посредством сравнения интенсивностей флуоресцентных сигналов элементов, в которых образовались совершенные и несовершенные гибридизационные комплексы. Сравнение флуоресцентных сигналов элементов проводят внутри каждой из групп 1-4 (Таблица 2, Фигура 2). Максимальный сигнал элемента в группе свидетельствовует о наличии или отсутствии однонуклеотидного полиморфизма С2611Т в каждой отдельной копии анализируемого гена 23S рРНК N. gonorrhoeae.
Полученные после проведения анализа гибридизационные картины приведены на Фигурах 3-5. На Фигуре 3 представлен результат анализа ДНК изолята с МПКазм=0,12 мг/л. Сигналы всех элементов биочипа, содержащих зонды дикого типа, превосходят сигналы элементов, содержащих зонды с заменой С2611Т, соответствующей искомому полиморфизму. Все копии гена 23 S рРНК относятся к дикому типу по положению 2611, а сам изолят является чувствительным к азитромицину, что подтверждено результатами анализа на биочипе.
На Фигуре 4 представлен результат анализа ДНК изолята с МПКазм=2 мг/л. Как свидетельствует гибридизационная картина, в группах элементов 1 и 3, сигналы элементов, содержащих зонды с заменой С2611Т, превосходят сигналы элементов, содержащих зонды дикого типа, тем самым геномная ДНК изолята содержит полиморфизм С2611Т в первой и третьей копии гена 23S рРНК. Таким образом, по результатам анализа на биочипе, данный изолят обладает устойчивостью к азитромицину.
На Фигуре 5 представлен результат анализа ДНК изолята с МПКазм=4 мг/л. Как свидетельствует гибридизационная картина, во всех группах сигнал элементов, содержащих зонды с заменой С2611Т, превосходят сигналы элементов, содержащих зонды дикого типа, тем самым геномная ДНК изолята содержит полиморфизм С2611Т во всех четырех копиях гена 23 S рРНК. Таким образом, по результатам анализа на биочипе, данный изолят обладает устойчивостью к азитромицину.
Корректность идентификации однонуклеотидного полиморфизма С2611Т в гене 23S рРНК, ассоциированного с устойчивостью N. gonorrhoeae к азитромицину, с использованием предлагаемого способа, проверяют методом секвенирования по Сэнгеру. Получают ПЦР-продукты каждой из четырех копий гена 23S рРНК N. gonorrhoeae и проводят их секвенирование, как описано ранее (Chisholm S.A., Dave J. et al. "High-level azithromycin resistance occurs in Neisseria gonorrhoeae as a result of a single point mutation in the 23S rRNA genes"; Antimicrob Agents Chemother. 2010, v. 54 (9), p. 3812-3816, doi:10.1128/AAC.00309-10). Результаты, полученные с использованием способа, предлагаемого в данном изобретении, и полученные методом секвенирования, совпали для всех анализируемых образцов.
Предлагаемый в данном изобретении способ идентификации однонуклеотидных полиморфизмов в многокопийном локусе генома методом петлевой ПЦР с последующей гибридизацией на биочипе позволяет быстро и точно идентифицировать полиморфизм в каждой отдельной копии исследуемого локуса, сокращая время проведения анализа.
Таблица 1

Таблица 2

--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="2022126923
Перечень последовательностей v.2.xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2023-08-07">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2022126923</ApplicationNumberText>
<FilingDate>2022-10-17</FilingDate>
</ApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное учреждение науки Институт молекулярной биологии им. В.А.
Энгельгардта Российской академии наук</ApplicantName>
<ApplicantNameLatin>Engelhardt Institute of Molecular Biology,
Russian Academy of Sciences</ApplicantNameLatin>
<InventionTitle languageCode="ru">СПОСОБ ИДЕНТИФИКАЦИИ
ОДНОНУКЛЕОТИДНЫХ ПОЛИМОРФИЗМОВ В МНОГОКОПИЙНОМ ЛОКУСЕ ГЕНОМА МЕТОДОМ
ПЕТЛЕВОЙ ПОЛИМЕРАЗНОЙ ЦЕПНОЙ РЕАКЦИИ С ПОСЛЕДУЮЩЕЙ ГИБРИДИЗАЦИЕЙ НА
БИОЛОГИЧЕСКОМ МИКРОЧИПЕ</InventionTitle>
<SequenceTotalQuantity>15</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>rRNA</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>23S rRNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q33">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>forward primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtgctgcgagcgctcatcacatcctggggctgtagtcgg</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>61</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..61</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>rRNA</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>23S rRNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>reverse primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atctccggtcggcgttggaagtttgagctatgccgtccacccgggtatc
cggggaggatta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>61</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..61</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>rRNA</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>23S rRNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q35">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>reverse primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atcagcactggccgttggaagtttgagctatgccgtccacctatcgtcg
atcccgttgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>61</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..61</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>rRNA</INSDFeature_key>
<INSDFeature_location>3</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>23S rRNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q36">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>reverse primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atcttgagagggcgttggaagtttgagctatgccgtccaccgttgcctt
ccacccttcgta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>61</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..61</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>rRNA</INSDFeature_key>
<INSDFeature_location>4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>23S rRNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q37">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>reverse primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atccgctgtaggcgttggaagtttgagctatgccgtccaccatggctac
cgccttcaacat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>13</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_structure</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q38">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>forward primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtgctgcgagcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_structure</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q45">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>reverse primer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtggacggcatagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>rRNA</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>allele</INSDQualifier_name>
<INSDQualifier_value>allele C, 23SrRNA gene, position 2611,
group 1, copy 1</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q31">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>rrnA (WT) C2611, oligonucleotide probe
for identification of C2611 SNP in copy 1 of the 23S rRNA gene, wild
type</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgaccggagatagggaccaaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>rRNA</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>allele</INSDQualifier_name>
<INSDQualifier_value>allele T, 23SrRNA gene, position 2611,
group 1, copy 1</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q32">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>rrnA C2611T, oligonucleotide probe for
identification of C2611T SNP in copy 1 of the 23S rRNA gene, T for C
substitution</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgaccggagatagagaccaaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>rRNA</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>allele</INSDQualifier_name>
<INSDQualifier_value>allele C, 23SrRNA gene, position 2611,
group 2, copy2</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q39">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>rrnB (WT) C2611, oligonucleotide probe
for identification of C2611 SNP in copy 2 of the 23S rRNA gene, wild
type</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcagtgctgatagggaccaaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>rRNA</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>allele</INSDQualifier_name>
<INSDQualifier_value>allele T, 23SrRNA gene, position 2611,
group 2, copy 2</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q40">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>rrnB C2611T, oligonucleotide probe for
identification of C2611T SNP in copy 2 of the 23S rRNA gene, T for C
substitution</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcagtgctgatagagaccaaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>rRNA</INSDFeature_key>
<INSDFeature_location>3</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>allele</INSDQualifier_name>
<INSDQualifier_value>allele C, 23SrRNA gene, position 2611,
group 3, copy 3</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q41">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>rrnC (WT) C2611, oligonucleotide probe
for identification of C2611 SNP in copy 3 of the 23S rRNA gene, wild
type</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctctcaagatagggaccaaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>rRNA</INSDFeature_key>
<INSDFeature_location>3</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>allele</INSDQualifier_name>
<INSDQualifier_value>allele T, 23SrRNA gene, position 2611,
group 3, copy 3</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q42">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>rrnC C2611T, oligonucleotide probe for
identification of C2611T SNP in copy 3 of the 23S rRNA gene, T for C
substitution</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctctcaagatagagaccaaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>rRNA</INSDFeature_key>
<INSDFeature_location>4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>allele</INSDQualifier_name>
<INSDQualifier_value>allele 3, 23SrRNA gene, position 2611,
group 4, copy 4</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q43">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>rrnD (WT) C2611, oligonucleotide probe
for identification of C2611 SNP in copy 4 of the 23S rRNA gene, wild
type</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctacagcggatagggaccaaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>rRNA</INSDFeature_key>
<INSDFeature_location>4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>allele</INSDQualifier_name>
<INSDQualifier_value>allele T, 23SrRNA gene, position 2611,
group 4, copy 4</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q44">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>rrnD C2611T, oligonucleotide probe for
identification of C2611T SNP in copy 4 of the 23S rRNA gene, T for C
substitution</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctacagcggatagagaccaaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБ ГЕНОТИПИРОВАНИЯ ИЗОЛЯТОВ NEISSERIA GONORRHOEAE НА БИОЛОГИЧЕСКОМ МИКРОЧИПЕ | 2023 |
|
RU2816767C1 |
| ОЛИГОНУКЛЕОТИДНЫЙ БИОЧИП ДЛЯ ИДЕНТИФИКАЦИИ ГЕНЕТИЧЕСКИХ ДЕТЕРМИНАНТ РЕЗИСТЕНТНОСТИ Neisseria gonorrhoeae К АНТИМИКРОБНЫМ ПРЕПАРАТАМ, НАБОР ОЛИГОНУКЛЕОТИДОВ, ИСПОЛЬЗУЕМЫХ ДЛЯ ИММОБИЛИЗАЦИИ НА БИОЧИПЕ | 2016 |
|
RU2636457C2 |
| Способ определения полиморфных маркеров в генах CYP2C19 и CYP2D6 для определения индивидуальной чувствительности к антидепрессантам | 2018 |
|
RU2716589C1 |
| СПОСОБ АНАЛИЗА ГЕНЕТИЧЕСКОГО ПОЛИМОРФИЗМА ДЛЯ ОПРЕДЕЛЕНИЯ ПРЕДРАСПОЛОЖЕННОСТИ К ШИЗОФРЕНИИ И АЛКОГОЛИЗМУ | 2012 |
|
RU2565036C2 |
| Способ анализа полиморфных маркеров в генах VKORC1, CYP4F2, CYP2C9, CYP2C19, ABCB1, ITGB3 для определения индивидуальной чувствительности к противосвертывающим препаратам | 2018 |
|
RU2689400C1 |
| НАБОР ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ И ЗОНДОВ ДЛЯ ГЕНОТИПИРОВАНИЯ ПОЛИМОРФНЫХ ЛОКУСОВ ДНК, АССОЦИИРОВАННЫХ С РИСКОМ РАЗВИТИЯ СПОРАДИЧЕСКОЙ ФОРМЫ БОЛЕЗНИ АЛЬЦГЕЙМЕРА В РОССИЙСКИХ ПОПУЛЯЦИЯХ | 2014 |
|
RU2600874C2 |
| Способ анализа полиморфных маркеров в генах метаболизма лекарственных препаратов и генах иммунного ответа при терапии острых лейкозов у детей | 2016 |
|
RU2643333C1 |
| Способ анализа терминальных мутаций в генах BRCA1, BRCA2, ATM и PALB2 с использованием мультиплексной ПЦР и последующей гибридизацией с олигонуклеотидным биологическим микрочипом (биочипом) | 2020 |
|
RU2729360C1 |
| Способ идентификации генетических полиморфизмов, влияющих на метаболизм противоопухолевых препаратов, с использованием биологических микрочипов | 2018 |
|
RU2697096C1 |
| СПОСОБ АНАЛИЗА СОМАТИЧЕСКИХ МУТАЦИЙ В ГЕНАХ EGFR, KRAS И BRAF С ИСПОЛЬЗОВАНИЕМ LNA-БЛОКИРУЮЩЕЙ МУЛЬТИПЛЕКСНОЙ ПЦР И ПОСЛЕДУЮЩЕЙ ГИБРИДИЗАЦИЕЙ С ОЛИГОНУКЛЕОТИДНЫМ БИОЛОГИЧЕСКИМ МИКРОЧИПОМ (БИОЧИПОМ) | 2014 |
|
RU2552483C1 |



Изобретение относится к области молекулярной биологии. Описан способ идентификации однонуклеотидных полиморфизмов в многокопийном локусе генома методом петлевой полимеразной цепной реакции с последующей гибридизацией на биологическом микрочипе. Способ включает несколько этапов амплификации, которые проводятся в одном реакционном объеме при разной температуре отжига праймеров, а также обеспечение биологического микрочипа с иммобилизованными олигонуклеотидными зондами, гибридизацию амплифицированных флуоресцентно-меченых продуктов на биологическом микрочипе, регистрацию и интерпретацию результатов гибридизации. Технический результат осуществления способа заключается в возможности проводить этапы амплификации в едином реакционном объеме, что избавляет от необходимости проведения нескольких последовательных стадий ПЦР с переносом продуктов реакции из одного объема в следующий. 2 з.п. ф-лы, 5 ил., 2 табл.
1. Способ идентификации однонуклеотидных полиморфизмов в многокопийном локусе генома методом петлевой полимеразной цепной реакции с последующей гибридизацией на биологическом микрочипе, включающий:
а) этап амплификации, в котором в качестве матрицы используют геномную ДНК, включающую многокопийный локус с исследуемым полиморфизмом и консервативным регионом, а в качестве праймеров используют один прямой праймер, последовательность которого состоит из двух частей - 3'-комплементарной последовательности фрагмента исследуемого локуса и 5'-адаптерной, последовательность которой некомплементарна последовательности исследуемого локуса, и набор обратных праймеров, последовательности которых состоят из двух частей - 3'-специфичной, комплементарной последовательности фрагмента, фланкирующего отдельную копию исследуемого локуса, и 5'-баркодирующей, комплементарной последовательности консервативного региона исследуемого локуса и содержащей уникальный для каждой копии исследуемого локуса набор однонуклеотидных замен - баркодов, тем самым в ходе этапа амплификации образуются ПЦР-продукты, ограниченные адаптерной последовательностью прямого праймера и баркодирующими последовательностями обратных праймеров;
б) этап амплификации, в котором в одной из цепей ПЦР-продукта, полученного на стадии (а), 5'-баркодирующая последовательность гибридизуется с комплементарной ей последовательностью консервативного региона исследуемого локуса, что обеспечивает образование и накопление продуктов с вторичной структурой типа петли с использованием праймеров стадии (а);
в) этап амплификации, в котором в качестве матрицы используют ПЦР-продукты с вторичной структурой типа петли, полученные на стадии (б), в качестве прямого праймера используют праймер, последовательность которого комплементарна адаптерной части прямого праймера, используемого на стадии (а), в качестве обратного праймера используют праймер, последовательность которого комплементарна не содержащему баркодов фрагменту баркодирующей части обратного праймера, используемого на стадии (а), при этом концентрации прямого и обратного праймеров в реакционной смеси могут различаться в 10 и более раз, а в реакционную смесь добавляют флуоресцентно-меченый дезоксиуридинтрифосфат с целью получения преимущественно одноцепочечных флуоресцентно-меченых ПЦР-продуктов, включающих последовательность локуса с однонуклеотидным полиморфизмом и последовательность баркодирующей части, соответствующую отдельной копии исследуемого локуса;
г) обеспечение биологического микрочипа для идентификации однонуклеотидного полиморфизма в исследуемом локусе, представляющего собой матрицу элементов, в каждом из которых иммобилизован уникальный олигонуклеотидный зонд, имеющий последовательность, комплементарную последовательности одноцепочечного фрагмента, полученного на стадии (в), и выбранную из группы, включающей: i) последовательности локуса дикого типа и фрагмента баркодирующей части, соответствующей отдельной копии исследуемого локуса; ii) последовательности локуса с заменой, характеризующей однонуклеотидный полиморфизм, и фрагмента соответствующей отдельной копии исследуемого локуса;
д) гибридизацию амплифицированных флуоресцентно-меченых ПЦР-продуктов, полученных на стадии (в), на биологическом микрочипе, полученном на стадии (г), с образованием совершенных и несовершенных дуплексов с иммобилизованными олигонуклеотидными зондами;
е) регистрацию результатов гибридизации на биологическом микрочипе, проведенной на стадии (д) с использованием портативного анализатора флуоресценции и программного обеспечения, что позволяет использовать программную обработку интенсивностей сигналов с последующей интерпретацией результатов.
2. Способ по п. 1, характеризующийся тем, что разница в температурах отжига праймеров, используемых на стадиях (а) и (в), составляет не менее 10°С, что позволяет проводить этапы амплификации (а)-(в) в едином реакционном объеме последовательно при разных температурах отжига.
3. Способ по п. 1, характеризующийся тем, что на стадии (е) совершенные дуплексы регистрируют в элементах биологического микрочипа в зависимости от последовательности баркодирующей части, что позволяет идентифицировать однонуклеотидный полиморфизм в каждой отдельной копии исследуемого локуса.
Авторы
Даты
2023-12-07—Публикация
2022-10-17—Подача