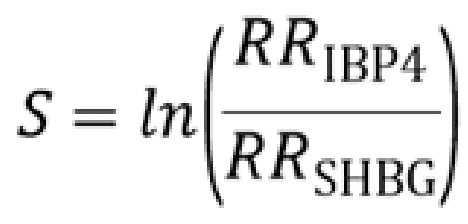[0001] По данной заявке испрашивают преимущество приоритета предварительной заявки США № 62/290,796, поданной 3 февраля 2016 года, предварительной заявки США № 62/387,420, поданной 24 декабря 2015 года, и предварительной заявки США № 62/182,349, поданной 19 июня 2015 года, полное содержание каждой из которых включено в настоящее описание посредством ссылки.
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
[0001.1] Данная заявка содержит список последовательностей, который подан в электронной форме в формате ASCII и, таким образом, включен по ссылке во всей своей полноте. Указанная ASCII копия, созданная 12 августа 2016 года, носит название 13271-018-228_SL.txt и имеет размер 31211 байтов.
[0002] Изобретение в целом относится к области репродуктивной медицины и, более конкретно, к композициям и способам для определения вероятности преждевременных родов у беременной женщины.
ПРЕДПОСЫЛКИ
[0003] Согласно Всемирной организации здравоохранения, каждый год преждевременно рождается оценочно 15 миллионов детей (до 37 полных недель беременности). Почти во всех странах с достоверными данными, частота преждевременных родов растет. См., World Health Organization; March of Dimes; The Partnership for Maternal, Newborn & Child Health; Save the Children, Born too soon: the global action report on preterm birth, ISBN 9789241503433(2012). Оценочно 1 миллион детей погибают ежегодно от осложнений преждевременных родов. Во всем мире преждевременные роды являются ведущей причиной смерти новорожденных (детей в первые четыре недели жизни) и второй ведущей причиной смерти после пневмонии у детей в возрасте до пяти лет. Многие выжившие сталкиваются с пожизненной нетрудоспособностью, включая затруднения при обучении и проблемы со слухом и зрением.
[0004] В 184 странах с достоверными данными, частота преждевременных родов находится в диапазоне от 5% до 18% родившихся детей. Blencowe et al., «National, regional and worldwide estimates of preterm birth» The Lancet, 9; 379(9832):2162-72 (2012). Хотя более 60% преждевременных родов происходит в Африке и Южной Азии, преждевременные роды, тем не менее, представляют собой глобальную проблему. Страны с наибольшим числом включают Бразилию, Индию, Нигерию и Соединенные Штаты Америки. Из 11 стран с частотой преждевременных родов выше 15%, все, кроме двух, находятся в странах Африки южнее Сахары. В беднейших странах усредненно 12% детей рождаются слишком рано по сравнению с 9% в странах с более высоким доходом. В пределах определенной страны более бедные семьи имеют более высокий риск. Больше трех четвертей преждевременно рожденных детей можно спасти, используя оправданный, экономически эффективный подход, например, предродовые инъекции стероидов, которые делают беременным женщинам с риском преждевременных сокращений матки, чтобы стабилизировать легкие ребенка.
[0005] Младенцы, рожденные преждевременно, имеют более высокий риск, чем младенцы, рожденные в срок, в отношении смертности и различных проблем со здоровьем и развитием. Осложнения включают острые проблемы с дыхательной, пищеварительной, иммунной, центральной нервной системой, слухом и зрением, а также отсроченные моторные, когнитивные проблемы, проблемы со слухом и зрением, поведенческие, социально-эмоциональные проблемы, проблемы со здоровьем и ростом. Преждевременные роды младенца также могут повлечь существенное эмоциональное и экономическое бремя для семей, а также оказывают влияние на службы общественного сектора, такие как медицинское страхование, система образования и другие системы социальной поддержки. Наибольшему риску смертности и распространенности подвержены те младенцы, которые рождены на самых ранних сроках беременности. Однако младенцы, рожденные ближе к сроку, составляют наибольшее число младенцев, рожденных преждевременно, и также испытывают больше осложнений, чем младенцы, рожденные в срок.
[0006] Для того чтобы предотвращать преждевременные роды у женщин, которые беременны меньше 24 недель и у которых ультразвук показывает раскрытие шейки матки, можно использовать хирургическую процедуру, известную как серкляж шейки матки, при которой шейку матки прошивают прочной нитью. Для женщин, беременных меньше 34 недель, с активными преждевременными сокращениями матки может быть необходимой госпитализация, а также введение лекарственных средств для того, чтобы временно остановить преждевременные сокращения матки и/или способствовать развитию легких плода. Если у беременных женщин определяют риск преждевременных родов, поставщики медицинских услуг могут реализовать различные клинические стратегии, которые могут включать превентивное введение лекарственных средств, например, инъекции 17-α гидроксипрогестерона капроата (Makena) и/или вагинальный гель с прогестероном, цервикальные пессарии, ограничение половой активности и/или других физических активностей и изменение лечения хронических состояний, таких как диабет и высокое кровяное давление, которые увеличивают риск преждевременных сокращений матки.
[0007] Существует высокая потребность в том, чтобы идентифицировать женщин с риском преждевременных родов и предоставлять им надлежащий предродовой уход. Женщинам, у которых идентифицирован высокий риск, можно назначать более интенсивное предродовое наблюдение и профилактические вмешательства. Существующие стратегии оценки риска основаны на акушерском и медицинском анамнезе, а также клиническом осмотре, но эти стратегии способны идентифицировать только небольшую процентную долю женщин, которые имеют риск преждевременного родоразрешения. Предшествующий анамнез самопроизвольных PTB (sPTB) в настоящее время является единственным сильным предиктором последующих PTB. После одних предыдущих sPTB вероятность вторых PTB составляет 30-50%. Другие материнские факторы риска включают: черную расу, низкий материнский индекс массы тела и малую длину шейки матки. Исследования биологических маркеров амниотической жидкости, цервиковагинальной жидкости и сыворотки для того, чтобы предсказать sPTB, показывают, что множество молекулярных путей нарушено у женщин, которые в конечном итоге рожают преждевременно. Надежная ранняя идентификация риска преждевременных родов сделает возможным планирование подходящего мониторинга и клинического ведения для предотвращения преждевременного родоразрешения. Такой мониторинг и ведение могут включать: более частые визиты для наблюдения за беременностью, последовательные измерения длины шейки матки, расширенное обучение в отношении признаков и симптомов ранних преждевременных сокращений матки, вмешательства в образ жизни для изменения рискованного поведения, такого как прекращение курения, цервикальные пессарии и лечение прогестероном. Наконец, надежная предродовая идентификация риска преждевременных родов также является ключевой для экономически эффективного размещения ресурсов мониторинга.
[0008] Несмотря на интенсивные исследования для того, чтобы идентифицировать женщин с риском, алгоритмы предсказания PTB, основанные лишь на клинических и демографических факторах или использующие измеренные сывороточные или вагинальные биологические маркеры, не привели к клинически эффективным тестам. Более точные способы идентификации женщин с риском во время их первой беременности и на достаточно ранних сроках беременности необходимы для того, чтобы сделать возможным клиническое вмешательство. Настоящее изобретение направлено на эту потребность посредством предоставления композиций и способов для определения того, имеет ли беременная женщина риск преждевременных родов. Также предоставлены связанные преимущества.
КРАТКОЕ ИЗЛОЖЕНИЕ
[0009] Настоящее изобретение предусматривает композиции и способы для предсказания вероятности преждевременных родов у беременной женщины.
[0010] Изобретение относится к выделенным биологическим маркерам, выбранным из группы, приведенной в таблице 26. Биологические маркеры по изобретению позволяют предсказывать риск преждевременных родов у беременной женщины. В некоторых вариантах осуществления выделенные биологические маркеры выбирают из группы, состоящей из IBP4, SHBG, PSG3, LYAM1, IGF2, CLUS, IBP3, INHBC, PSG2, PEDF, CD14 и APOC3.
[0011] Изобретение относится к суррогатным пептидам выделенных биологических маркеров, выбранных из группы, приведенной в таблице 26. В некоторых вариантах осуществления суррогатные пептиды выделенных биологических маркеров выбирают из группы суррогатных пептидов, приведенных в таблице 26. Биологические маркеры по изобретению и их суррогатные пептиды можно использовать в способах предсказания риска преждевременных родов у беременной женщины. В некоторых вариантах осуществления суррогатные пептиды соответствуют выделенным биологическим маркерам, выбранным из группы, состоящей из IBP4, SHBG, PSG3, LYAM1, IGF2, CLUS, IBP3, INHBC, PSG2, PEDF, CD14 и APOC3.
[0012] Изобретение относится к меченным стабильными изотопами стандартным пептидам (SIS пептидам), соответствующим суррогатным пептидам, выбранным из группы, приведенной в таблице 26. Биологические маркеры по изобретению, их суррогатные пептиды и SIS пептиды можно использовать в способах предсказания риска преждевременных родов у беременной женщины. В некоторых вариантах осуществления SIS пептиды соответствуют суррогатным пептидам выделенных биологических маркеров, выбранных из группы, состоящей из IBP4, SHBG, PSG3, LYAM1, IGF2, CLUS, IBP3, INHBC, PSG2, PEDF, CD14 и APOC3.
[0013] Изобретение относится к паре выделенных биологических маркеров, выбранной из группы, состоящей из выделенных биологических маркеров, перечисленных в таблице 26, где пара биологических маркеров демонстрирует изменение значения соотношения между беременными женщинами с риском преждевременных родов и полносрочными контролями.
[0014] Изобретение относится к паре выделенных биологических маркеров, выбранных из группы, состоящей из IBP4/SHBG, VTNC/VTDB, VTNC/SHBG, CATD/SHBG, PSG2/ITIH4, CHL1/ITIH4, PSG2/C1QB, PSG2/FBLN3, HPX/IBP4 и HPX/PTGDS, где пара биологических маркеров демонстрирует изменение значения соотношения между беременными женщинами с риском преждевременных родов и полносрочными контролями.
[0015] Изобретение относится к паре выделенных биологических маркеров, выбранных из группы, состоящей из IBP4/SHBG, IBP4/PSG3, IBP4/LYAM1, IBP4/IGF2, CLUS/IBP3, CLUS/IGF2, CLUS/LYAM1, INHBC/PSG3, INHBC/IGF2, PSG2/LYAM1, PSG2/IGF2, PSG2/LYAM1, PEDF/PSG3, PEDF/SHBG, PEDF/LYAM1, CD14/LYAM1 и APOC3/LYAM1, где пара биологических маркеров демонстрирует изменение значения соотношения между беременными женщинами с риском преждевременных родов и полносрочными контролями.
[0016] В одном из вариантов осуществления изобретение относится к паре выделенных биологических маркеров, выбранной из группы, состоящей из IBP4/SHBG, VTNC/VTDB, VTNC/SHBG, CATD/SHBG, PSG2/ITIH4, CHL1/ITIH4, PSG2/C1QB, PSG2/FBLN3, HPX/IBP4 и HPX/PTGDS, где пара биологических маркеров демонстрирует изменение значения обращения между беременными женщинами с риском преждевременных родов и полносрочными контролями.
[0017] В одном из вариантов осуществления изобретение относится к паре выделенных биологических маркеров, выбранных из группы, состоящей из IBP4/SHBG, IBP4/PSG3, IBP4/LYAM1, IBP4/IGF2, CLUS/IBP3, CLUS/IGF2, CLUS/LYAM1, INHBC/PSG3, INHBC/IGF2, PSG2/LYAM1, PSG2/IGF2, PSG2/LYAM1, PEDF/PSG3, PEDF/SHBG, PEDF/LYAM1, CD14/LYAM1 и APOC3/LYAM1, где пара биологических маркеров демонстрирует изменение значения обращения между беременными женщинами с риском преждевременных родов и полносрочными контролями.
[0018] В одном из вариантов осуществления изобретение относится к композиции, содержащей пару суррогатных пептидов, соответствующую паре биологических маркеров, выбранной из группы, состоящей из IBP4/SHBG, VTNC/VTDB, VTNC/SHBG, CATD/SHBG, PSG2/ITIH4, CHL1/ITIH4, PSG2/C1QB, PSG2/FBLN3, HPX/IBP4 и HPX/PTGDS, где пара биологических маркеров демонстрирует изменение значения обращения между беременными женщинами с риском преждевременных родов и полносрочными контролями. В одном из вариантов осуществления композиция содержит меченные стабильными изотопами стандартные пептиды (SIS пептиды) для каждого из суррогатных пептидов.
[0019] В одном из вариантов осуществления изобретение относится к композиции, содержащей пару суррогатных пептидов, соответствующую паре биологических маркеров, выбранных из группы, состоящей из IBP4/SHBG, IBP4/PSG3, IBP4/LYAM1, IBP4/IGF2, CLUS/IBP3, CLUS/IGF2, CLUS/LYAM1, INHBC/PSG3, INHBC/IGF2, PSG2/LYAM1, PSG2/IGF2, PSG2/LYAM1, PEDF/PSG3, PEDF/SHBG, PEDF/LYAM1, CD14/LYAM1 и APOC3/LYAM1, где пара биологических маркеров демонстрирует изменение значения обращения между беременными женщинами с риском преждевременных родов и полносрочными контролями. В одном из вариантов осуществления композиция содержит меченные стабильными изотопами стандартные пептиды (SIS пептиды) для каждого из суррогатных пептидов.
[0020] В конкретном варианте осуществления изобретение относится к паре выделенных биологических маркеров IBP4/SHBG, где пара биологических маркеров демонстрирует изменение значения обращения между беременными женщинами с риском преждевременных родов по сравнению с полносрочными контролями. В дополнительном варианте осуществления изобретение относится к паре выделенных биологических маркеров IBP4/SHBG, где пара биологических маркеров демонстрирует более высокое соотношение у беременных женщин с риском преждевременных родов по сравнению с полносрочными контролями.
[0021] В одном из вариантов осуществления изобретение относится к композиции, содержащей пару суррогатных пептидов, соответствующую паре биологических маркеров IBP4/SHBG, где пара биологических маркеров демонстрирует более высокое соотношение у беременных женщин с риском преждевременных родов по сравнению с полносрочными контролями. В одном из вариантов осуществления композиция содержит меченные стабильными изотопами стандартные пептиды (SIS пептиды) для каждого из суррогатных пептидов.
[0022] В дополнительном варианте осуществления изобретение относится к панели из по меньшей мере двух пар биологических маркеров, выбранных из группы, состоящей из IBP4/SHBG, VTNC/VTDB, VTNC/SHBG, CATD/SHBG, PSG2/ITIH4, CHL1/ITIH4, PSG2/C1QB, PSG2/FBLN3, HPX/IBP4 и HPX/PTGDS, где каждая из пар демонстрирует изменение значения обращения между беременными женщинами с риском преждевременных родов и полносрочными контролями. В одном из вариантов осуществления панель содержит меченные стабильными изотопами стандартные пептиды (SIS пептиды) для суррогатных пептидов, полученных из каждого из указанных биологических маркеров.
[0023] В дополнительном варианте осуществления изобретение относится к панели из по меньшей мере двух пар биологических маркеров, выбранных из группы, состоящей из IBP4/SHBG, IBP4/PSG3, IBP4/LYAM1, IBP4/IGF2, CLUS/IBP3, CLUS/IGF2, CLUS/LYAM1, INHBC/PSG3, INHBC/IGF2, PSG2/LYAM1, PSG2/IGF2, PSG2/LYAM1, PEDF/PSG3, PEDF/SHBG, PEDF/LYAM1, CD14/LYAM1 и APOC3/LYAM1, где каждая из пар демонстрирует изменение значения обращения между беременными женщинами с риском преждевременных родов и полносрочными контролями. В одном из вариантов осуществления панель содержит меченные стабильными изотопами стандартные пептиды (SIS пептиды) для суррогатных пептидов, полученных из каждого из указанных биологических маркеров.
[0024] В дополнительном варианте осуществления изобретение относится к панели из по меньшей мере двух пар суррогатных пептидов, каждая пара суррогатных пептидов соответствует паре биологических маркеров, выбранной из группы, состоящей из IBP4/SHBG, VTNC/VTDB, VTNC/SHBG, CATD/SHBG, PSG2/ITIH4, CHL1/ITIH4, PSG2/C1QB, PSG2/FBLN3, HPX/IBP4 и HPX/PTGDS, где каждая из пар демонстрирует изменение значения обращения между беременными женщинами с риском преждевременных родов и полносрочными контролями. В одном из вариантов осуществления панель содержит меченные стабильными изотопами стандартные пептиды (SIS пептиды) для каждого из суррогатных пептидов.
[0025] В дополнительном варианте осуществления изобретение относится к панели из по меньшей мере двух пар суррогатных пептидов, каждая пара суррогатных пептидов соответствует паре биологических маркеров, выбранной из группы, состоящей из IBP4/SHBG, IBP4/PSG3, IBP4/LYAM1, IBP4/IGF2, CLUS/IBP3, CLUS/IGF2, CLUS/LYAM1, INHBC/PSG3, INHBC/IGF2, PSG2/LYAM1, PSG2/IGF2, PSG2/LYAM1, PEDF/PSG3, PEDF/SHBG, PEDF/LYAM1, CD14/LYAM1 и APOC3/LYAM1, где каждая из пар демонстрирует изменение значения обращения между беременными женщинами с риском преждевременных родов и полносрочными контролями. В одном из вариантов осуществления панель содержит меченные стабильными изотопами стандартные пептиды (SIS пептиды) для каждого из суррогатных пептидов.
[0026] В дополнительном варианте осуществления изобретение относится к панели из по меньшей мере двух пар суррогатных пептидов, каждая пара суррогатных пептидов соответствует паре биологических маркеров, выбранной из группы, состоящей из IBP4/SHBG, VTNC/VTDB, VTNC/SHBG, CATD/SHBG, PSG2/ITIH4, CHL1/ITIH4, PSG2/C1QB, PSG2/FBLN3, HPX/IBP4 и HPX/PTGDS, где по меньшей мере одна из пар демонстрирует изменение значения обращения между беременными женщинами с риском преждевременных родов и полносрочными контролями. В одном из вариантов осуществления композиция содержит меченные стабильными изотопами стандартные пептиды (SIS пептиды) для каждого из суррогатных пептидов.
[0027] В дополнительном варианте осуществления изобретение относится к панели из по меньшей мере двух пар суррогатных пептидов, каждая пара суррогатных пептидов соответствует паре биологических маркеров, выбранной из группы, состоящей из IBP4/SHBG, IBP4/PSG3, IBP4/LYAM1, IBP4/IGF2, CLUS/IBP3, CLUS/IGF2, CLUS/LYAM1, INHBC/PSG3, INHBC/IGF2, PSG2/LYAM1, PSG2/IGF2, PSG2/LYAM1, PEDF/PSG3, PEDF/SHBG, PEDF/LYAM1, CD14/LYAM1 и APOC3/LYAM1, где по меньшей мере одна из пар демонстрирует изменение значения обращения между беременными женщинами с риском преждевременных родов и полносрочными контролями. В одном из вариантов осуществления композиция содержит меченные стабильными изотопами стандартные пептиды (SIS пептиды) для каждого из суррогатных пептидов.
[0028] В дополнительном варианте осуществления изобретение относится к панели из по меньшей мере двух пар суррогатных пептидов, каждая пара суррогатных пептидов соответствует паре биологических маркеров, выбранной из группы, состоящей из IBP4/SHBG, VTNC/VTDB, VTNC/SHBG, CATD/SHBG, PSG2/ITIH4, CHL1/ITIH4, PSG2/C1QB, PSG2/FBLN3, HPX/IBP4 и HPX/PTGDS, где вычисленная оценка, полученная для панели из по меньшей мере двух пар биологических маркеров, демонстрирует изменение значения между беременными женщинами и полносрочными контролями. В одном из вариантов осуществления композиция содержит меченные стабильными изотопами стандартные пептиды (SIS пептиды) для каждого из суррогатных пептидов.
[0029] В дополнительном варианте осуществления изобретение относится к панели из по меньшей мере двух пар суррогатных пептидов, каждая пара суррогатных пептидов соответствует паре биологических маркеров, выбранной из группы, состоящей из IBP4/SHBG, IBP4/PSG3, IBP4/LYAM1, IBP4/IGF2, CLUS/IBP3, CLUS/IGF2, CLUS/LYAM1, INHBC/PSG3, INHBC/IGF2, PSG2/LYAM1, PSG2/IGF2, PSG2/LYAM1, PEDF/PSG3, PEDF/SHBG, PEDF/LYAM1, CD14/LYAM1 и APOC3/LYAM1, где вычисленная оценка, полученная для панели из по меньшей мере двух пар биологических маркеров, демонстрирует изменение значения между беременными женщинами и полносрочными контролями. В одном из вариантов осуществления композиция содержит меченные стабильными изотопами стандартные пептиды (SIS пептиды) для каждого из суррогатных пептидов.
[0030] В одном из вариантов осуществления изобретение относится к способу определения вероятности преждевременных родов у беременной женщины, способ включает измерение в биологическом образце, полученном от беременной женщины, соотношения для по меньшей мере одной пары биологических маркеров, выбранной из группы, состоящей из IBP4/SHBG, VTNC/VTDB, VTNC/SHBG, CATD/SHBG, PSG2/ITIH4, CHL1/ITIH4, PSG2/C1QB, PSG2/FBLN3, HPX/IBP4 и HPX/PTGDS, чтобы определять вероятность преждевременных родов у беременной женщины. В некоторых вариантах осуществления беременная женщина имеет индекс массы тела (BMI) больше 22 и меньше или равный 37 кг/м2.
[0031] В одном из вариантов осуществления изобретение относится к способу определения вероятности преждевременных родов у беременной женщины, способ включает измерение в биологическом образце, полученном от беременной женщины, соотношения для по меньшей мере одной пары биологических маркеров, выбранной из группы, состоящей из IBP4/SHBG, IBP4/PSG3, IBP4/LYAM1, IBP4/IGF2, CLUS/IBP3, CLUS/IGF2, CLUS/LYAM1, INHBC/PSG3, INHBC/IGF2, PSG2/LYAM1, PSG2/IGF2, PSG2/LYAM1, PEDF/PSG3, PEDF/SHBG, PEDF/LYAM1, CD14/LYAM1 и APOC3/LYAM1, чтобы определять вероятность преждевременных родов у беременной женщины. В некоторых вариантах осуществления беременная женщина имеет индекс массы тела (BMI) больше 22 и меньше или равный 37 кг/м2. В некоторых вариантах осуществления способ включает начальную стадию получения биологического образца. В некоторых вариантах осуществления способ включает обнаружение, измерение или количественное определение SIS суррогатного пептид каждого из биологических маркеров.
[0032] В некоторых вариантах осуществления определение вероятности преждевременных родов у беременной женщины включает начальную стадию, которая включает формирование показателя вероятности/риска посредством измерения соотношения выделенных биологических маркеров, выбранных из группы в когорте преждевременных беременностей и полносрочных беременностей с известным гестационным возрастом при родах. В дополнительных вариантах осуществления показатель преждевременного риска формируют посредством измерения соотношения IBP4/SHBG в когорте преждевременных и полносрочных беременностей, где зарегистрирован гестационный возраст при рождении. В некоторых вариантах осуществления определение вероятности преждевременных родов у беременной женщины включает измерение соотношения IBP4/SHBG и сравнение значения с показателем, чтобы получать преждевременный риск, используя те же технологии выделения и измерения, что и в основной группе, чтобы извлекать IBP4/SHBG.
[0033] В одном из вариантов осуществления изобретение относится к способу определения вероятности преждевременных родов у беременной женщины, способ включает измерение в биологическом образце, полученном от беременной женщины, значения обращения для по меньшей мере одной пары биологических маркеров, выбранной из группы, состоящей из IBP4/SHBG, VTNC/VTDB, VTNC/SHBG, CATD/SHBG, PSG2/ITIH4, CHL1/ITIH4, PSG2/C1QB, PSG2/FBLN3, HPX/IBP4 и HPX/PTGDS, чтобы определять вероятность преждевременных родов у беременной женщины. В некоторых вариантах осуществления беременная женщина имеет индекс массы тела (BMI) больше 22 и меньше или равный 37 кг/м2. В некоторых вариантах осуществления способ включает начальную стадию получения биологического образца. В некоторых вариантах осуществления способ включает обнаружение, измерение или количественное определение SIS суррогатного пептида каждого из биологических маркеров.
[0034] В одном из вариантов осуществления изобретение относится к способу определения вероятности преждевременных родов у беременной женщины, способ включает измерение в биологическом образце, полученном от беременной женщины, значения обращения для по меньшей мере одной пары биологических маркеров, выбранной из группы, состоящей из IBP4/SHBG, IBP4/PSG3, IBP4/LYAM1, IBP4/IGF2, CLUS/IBP3, CLUS/IGF2, CLUS/LYAM1, INHBC/PSG3, INHBC/IGF2, PSG2/LYAM1, PSG2/IGF2, PSG2/LYAM1, PEDF/PSG3, PEDF/SHBG, PEDF/LYAM1, CD14/LYAM1 и APOC3/LYAM1, чтобы определять вероятность преждевременных родов у беременной женщины. В некоторых вариантах осуществления беременная женщина имеет индекс массы тела (BMI) больше 22 и меньше или равный 37 кг/м2. В некоторых вариантах осуществления способ включает начальную стадию получения биологического образца. В некоторых вариантах осуществления способ включает обнаружение, измерение или количественное определение SIS суррогатного пептида каждого из биологических маркеров.
[0035] В другом варианте осуществления изобретение относится к способу определения вероятности преждевременных родов у беременной женщины, способ включает измерение в биологическом образце, полученном от беременной женщины, изменения значения обращения для панели из по меньшей мере двух пар биологических маркеров, выбранных из группы, состоящей из IBP4/SHBG, VTNC/VTDB, VTNC/SHBG, CATD/SHBG, PSG2/ITIH4, CHL1/ITIH4, PSG2/C1QB, PSG2/FBLN3, HPX/IBP4 и HPX/PTGDS, чтобы определять вероятность преждевременных родов у беременной женщины. В другом варианте осуществления изобретение относится к способу определения вероятности преждевременных родов у беременной женщины, способ включает измерение в биологическом образце, полученном от беременной женщины, изменения значения обращения для панели из по меньшей мере двух пар биологических маркеров, выбранных из группы, состоящей из IBP4/SHBG, IBP4/PSG3, IBP4/LYAM1, IBP4/IGF2, CLUS/IBP3, CLUS/IGF2, CLUS/LYAM1, INHBC/PSG3, INHBC/IGF2, PSG2/LYAM1, PSG2/IGF2, PSG2/LYAM1, PEDF/PSG3, PEDF/SHBG, PEDF/LYAM1, CD14/LYAM1 и APOC3/LYAM1, чтобы определять вероятность преждевременных родов у беременной женщины. В некоторых вариантах осуществления значение обращения раскрывает существование изменения относительных интенсивностей индивидуальных биологических маркеров между беременной женщиной и полносрочным контролем и отражает вероятность преждевременных родов у беременной женщины. В дополнительных вариантах осуществления стадия измерения включает измерение суррогатных пептидов биологических маркеров в биологическом образце, полученном от беременной женщины. В некоторых вариантах осуществления беременная женщина имеет индекс массы тела (BMI) больше 22 и меньше или равный 37 кг/м2. В некоторых вариантах осуществления способ включает начальную стадию получения биологического образца. В некоторых вариантах осуществления способ включает обнаружение, измерение или количественное определение SIS суррогатного пептида каждого из биологических маркеров.
[0036] В одном из вариантов осуществления изобретение относится к способу определения вероятности преждевременных родов у беременной женщины, способ включает измерение в биологическом образце, полученном от беременной женщины, значения обращения для пары биологических маркеров, состоящей из IBP4 и SHBG, чтобы определять вероятность преждевременных родов у беременной женщины. В некоторых вариантах осуществления беременная женщина имеет индекс массы тела (BMI) больше 22 и меньше или равный 37 кг/м2. В некоторых вариантах осуществления способ включает начальную стадию получения биологического образца. В некоторых вариантах осуществления способ включает обнаружение, измерение или количественное определение SIS суррогатного пептида каждого из биологических маркеров.
[0037] В одном из вариантов осуществления изобретение относится к способу определения вероятности преждевременных родов у беременной женщины, способ включает измерение в биологическом образце, полученном от беременной женщины, значения обращения для пары биологических маркеров, состоящего из соотношения IBP4 и SHBG (IBP4/SHBG), чтобы определять вероятность преждевременных родов у беременной женщины, где более высокое соотношение у беременной женщины по сравнению с полносрочными контролями отражает повышенный риск преждевременных родов. В дополнительных вариантах осуществления беременная женщина имеет индекс массы тела (BMI) больше 22 и меньше или равный 37 кг/м2. В некоторых вариантах осуществления способ включает начальную стадию получения биологического образца. В некоторых вариантах осуществления способ включает обнаружение, измерение или количественное определение SIS суррогатного пептида каждого из биологических маркеров.
[0038] В одном из вариантов осуществления изобретение относится к способу определения вероятности преждевременных родов у беременной женщины, способ включает измерение в биологическом образце, полученном от беременной женщины, значения обращения для пары биологических маркеров IBP4 и SHBG, чтобы определять вероятность преждевременных родов у беременной женщины. В некоторых вариантах осуществления беременная женщина имеет индекс массы тела (BMI) больше 22 и меньше или равный 37 кг/м2. В некоторых вариантах осуществления способ включает начальную стадию получения биологического образца. В некоторых вариантах осуществления способ включает обнаружение, измерение или количественное определение SIS суррогатного пептида каждого из биологических маркеров.
[0039] Изобретение также относится к способу обнаружения пары выделенных биологических маркеров, выбранной из группы, состоящей из IBP4/SHBG, IBP4/PSG3, IBP4/LYAM1, IBP4/IGF2, CLUS/IBP3, CLUS/IGF2, CLUS/LYAM1, INHBC/PSG3, INHBC/IGF2, PSG2/LYAM1, PSG2/IGF2, PSG2/LYAM1, PEDF/PSG3, PEDF/SHBG, PEDF/LYAM1, CD14/LYAM1 и APOC3/LYAM1, у беременной женщины, указанный способ включает стадии a. получения биологического образца от беременной женщины; b. обнаружения присутствия пары выделенных биологических маркеров в биологическом образце посредством контакта биологического образца с первым захватывающим средством, которое специфически связывает первый элемент указанной пары, и вторым захватывающим средством, которое специфически связывает второй элемент указанной пары; и обнаружения связывания между первым биологическим маркером указанной пары и первым захватывающим средством и между вторым элементом указанной пары и вторым захватывающим средством.
[0040] В одном из вариантов осуществления изобретение относится к способу обнаружения IBP4 и SHBG у беременной женщины, указанный способ включает стадии a. получения биологического образца от беременной женщины; b. обнаружения присутствия IBP4 и SHBG в биологическом образце посредством контакта биологического образца с захватывающим средством, которое специфически связывает IBP4, и захватывающим средством, которое специфически связывает SHBG; и c. обнаружения связывания между IBP4 и захватывающим средством и между SHBG и захватывающим средством. В одном из вариантов осуществления способ включает измерение значения обращения для пары биологических маркеров. В дополнительном варианте осуществления существование изменения значения обращения между беременной женщиной и полносрочным контролем отражает вероятность преждевременных родов у беременной женщины. В одном из вариантов осуществления образец получают между 19 и 21 неделями гестационного возраста. В дополнительном варианте осуществления захватывающее средство выбирают из группы, состоящей из антитела, фрагмента антитела, реактива на основе нуклеиновой кислоты, связывающего белок, низкомолекулярного соединения или его варианта. В дополнительном варианте осуществления способ осуществляют посредством анализа, выбранного из группы, состоящей из иммуноферментного анализа (EIA), твердофазного иммуноферментного анализа (ELISA) и радиоиммунного анализа (RIA).
[0041] Изобретение также относится к способу обнаружения пары выделенных биологических маркеров, выбранной из группы, состоящей из IBP4/SHBG, IBP4/PSG3, IBP4/LYAM1, IBP4/IGF2, CLUS/IBP3, CLUS/IGF2, CLUS/LYAM1, INHBC/PSG3, INHBC/IGF2, PSG2/LYAM1, PSG2/IGF2, PSG2/LYAM1, PEDF/PSG3, PEDF/SHBG, PEDF/LYAM1, CD14/LYAM1 и APOC3/LYAM1, у беременной женщины, указанный способ включает стадии a. получения биологического образца от беременной женщины; и b. обнаружения присутствия пары выделенных биологических маркеров в биологическом образце, включающего применение образца на протеомном технологическом маршруте, состоящем из масс-спектрометрического количественного определения.
[0042] В одном из вариантов осуществления изобретение относится к способу обнаружения IBP4 и SHBG у беременной женщины, указанный способ включает стадии a. получения биологического образца от беременной женщины; и b. обнаружения присутствия пары выделенных биологических маркеров в биологическом образце, включающего применение образца на протеомном технологическом маршруте, состоящем из масс-спектрометрического количественного определения.
[0043] В некоторых вариантах осуществления значение обращения раскрывает существование изменения относительных интенсивностей индивидуальных биологических маркеров между беременной женщиной и полносрочным контролем и отражает вероятность преждевременных родов у беременной женщины. В дополнительных вариантах осуществления стадия измерения включает измерение суррогатных пептидов биологических маркеров в биологическом образце, полученном от беременной женщины. В одном из вариантов осуществления показатель преждевременного риска формируют посредством измерения соотношения IBP4/SHBG в когорте преждевременных и полносрочных беременностей, где зарегистрирован гестационный возраст при рождении. Затем, в клинической практике измеряемое соотношение IBP4/SHBG при индивидуальной беременности сравнивают в виде показателя, чтобы получать преждевременный риск с использованием тех же технологий выделения и измерения, что и в основной группе, чтобы извлекать IBP4/SHBG.
[0044] Другие признаки и преимущества по изобретению видны из подробного описания и формулы изобретения.
КРАТКОЕ ОПИСАНИЕ ФИГУР
[0045] Фиг. 1. Интервалы взятия крови. Эффективность индивидуального обращения представлена для интервалов взятия крови. Представлены обращения: IBP4/SHBG; VTNC/VTDB; IBP4/SHBG; VTNC/SHBG; IBP4/SHBG; CATD/SHBG; PSG2/ITIH4; CHL1/ITIH4; PSG2/C1QB; PSG2/FBLN3; HEMO/IBP6; HEMO/PTGDS.
[0046] Фиг. 2. Исследуемый случай, верификационный случай и валидационный случай для GABD.
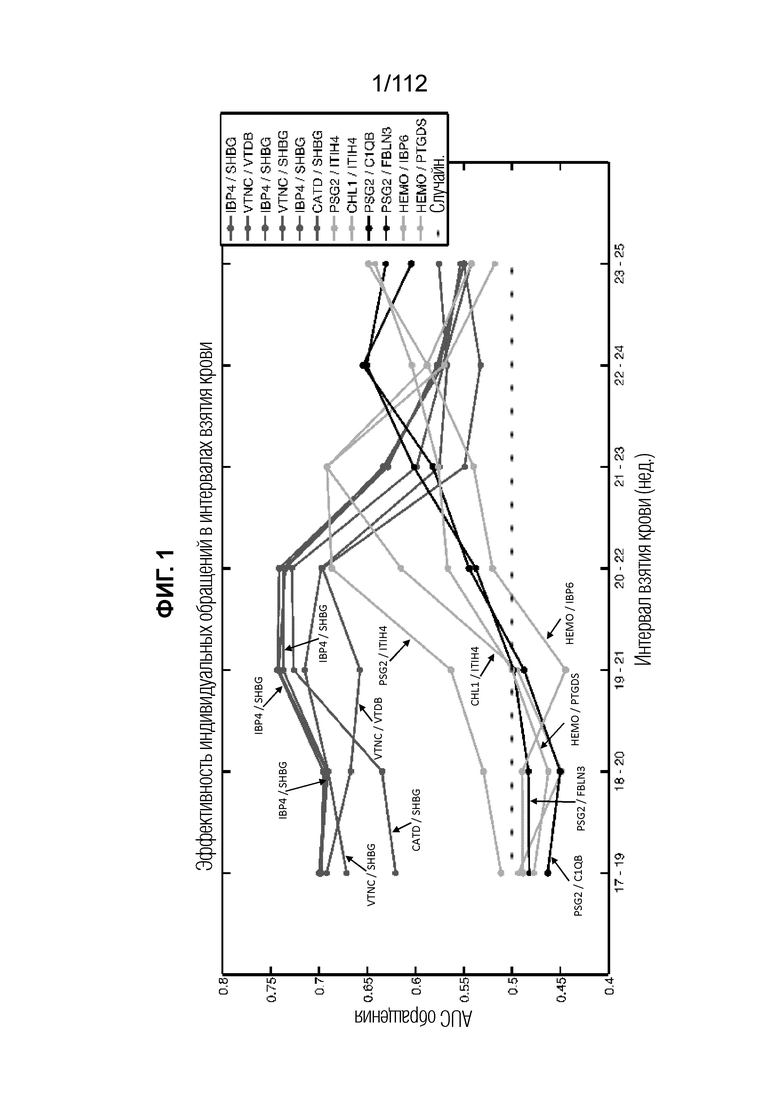
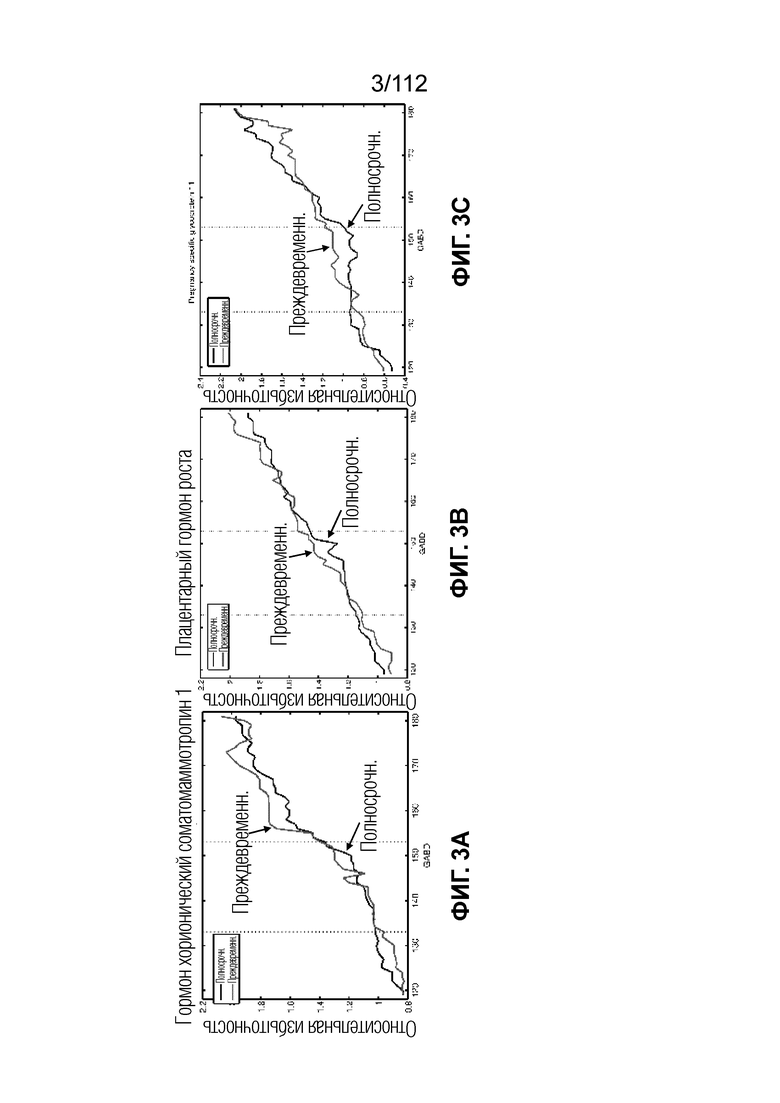
[0047] Фиг. 3. Экспрессия белков во время беременности. Различные белки можно анализировать на основании известного поведения белков и зная белки/пути, на которые не влияют преждевременные роды. На фиг. 3 представлена экспрессия белков, связанных с беременностью, во время беременности. На эти белки и их сети не влияет преждевременная патология в указанном гестационном возрасте.
[0048] Фиг. 4. Экспрессия белков во время беременности. На фиг. 4 представлена увеличенная версия графика, представленного на фиг. 3, который относится к плацентарному гормону роста.
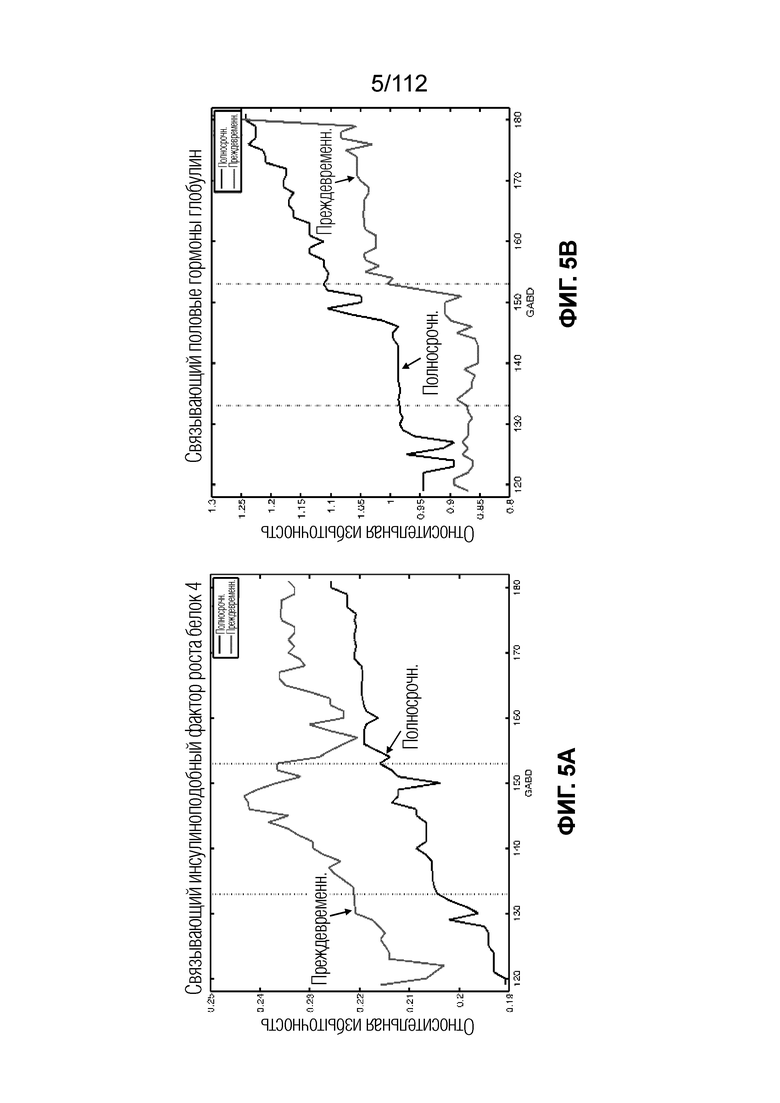
[0049] Фиг. 5. Белковая патология во время беременности. Связывающий инсулиноподобный фактор роста белок 4 (IBP4) сверхэкспрессирован по меньшей мере на 10% в интервале взятия крови 19-21 неделя. Связывающий половые гормоны глобулин (SHBG) недоэкспрессирован по меньшей мере на 10% в интервале взятия крови 19-21 неделя.
[0050] Фиг. 6. Критерии верификационного отбора. На фиг. 6 представлены критерии для осуществления клинически и аналитически надежного преждевременного теста высокой эффективности.
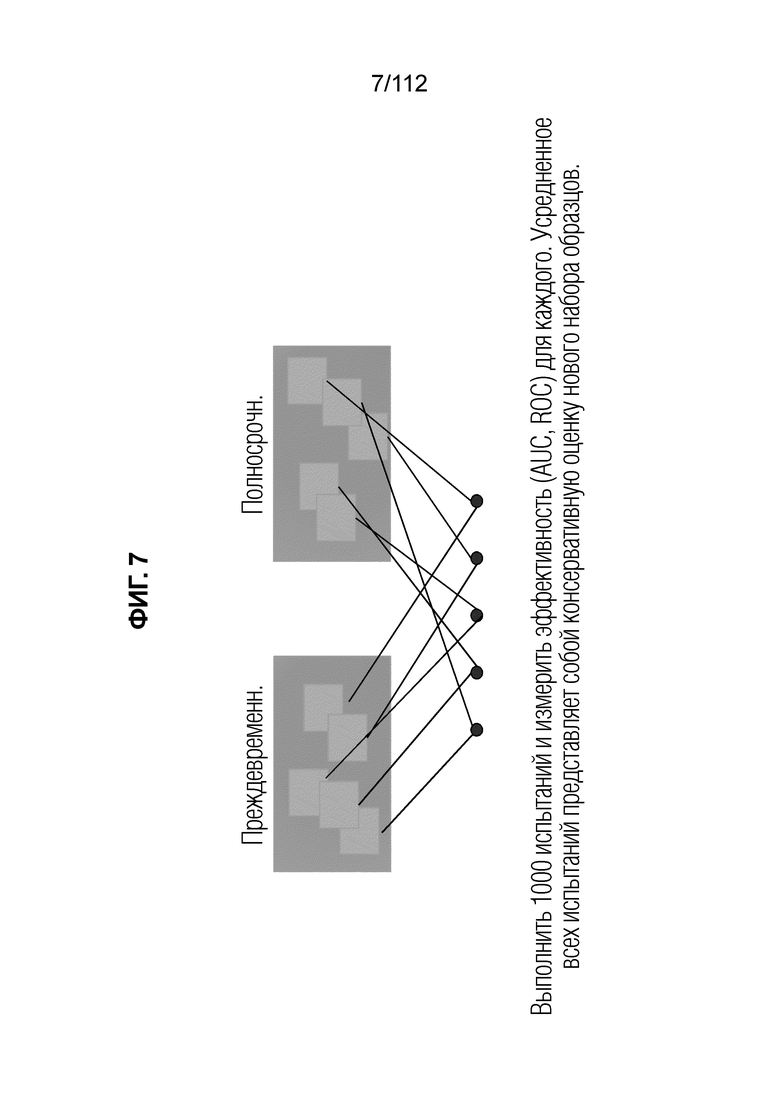
[0051] Фиг. 7. Перекрестная валидация способом Монте-Карло (MCVV). MCCV представляет собой консервативный способ, который оценивает, насколько хорошо классификатор работает на независимом наборе образцов, взятых в той же популяции (например, PAPR).
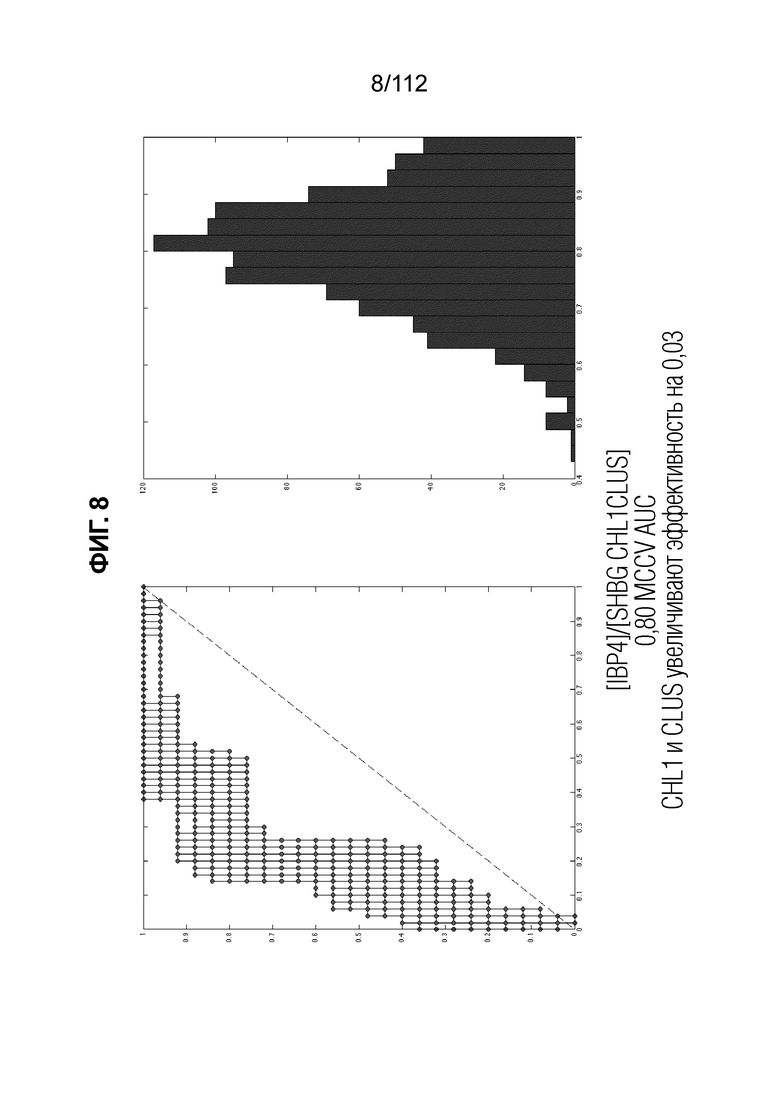
[0052] Фиг. 8. Анализ [IBP4]/ [SHBG CHL1 CLUS]. Эффективность CHL1 и CLUS, повышенная на 0,03 относительно только IBP4/SHBG.
[0053] Фиг. 9. Анализ мощности и размера образца. Анализ мощности и размера образца предсказывает вероятность того, что исследование имеет достаточную мощность, чтобы отклонять нуль-гипотезу (AUC=0,5) при порогах для числа образцов и оценок эффективности.
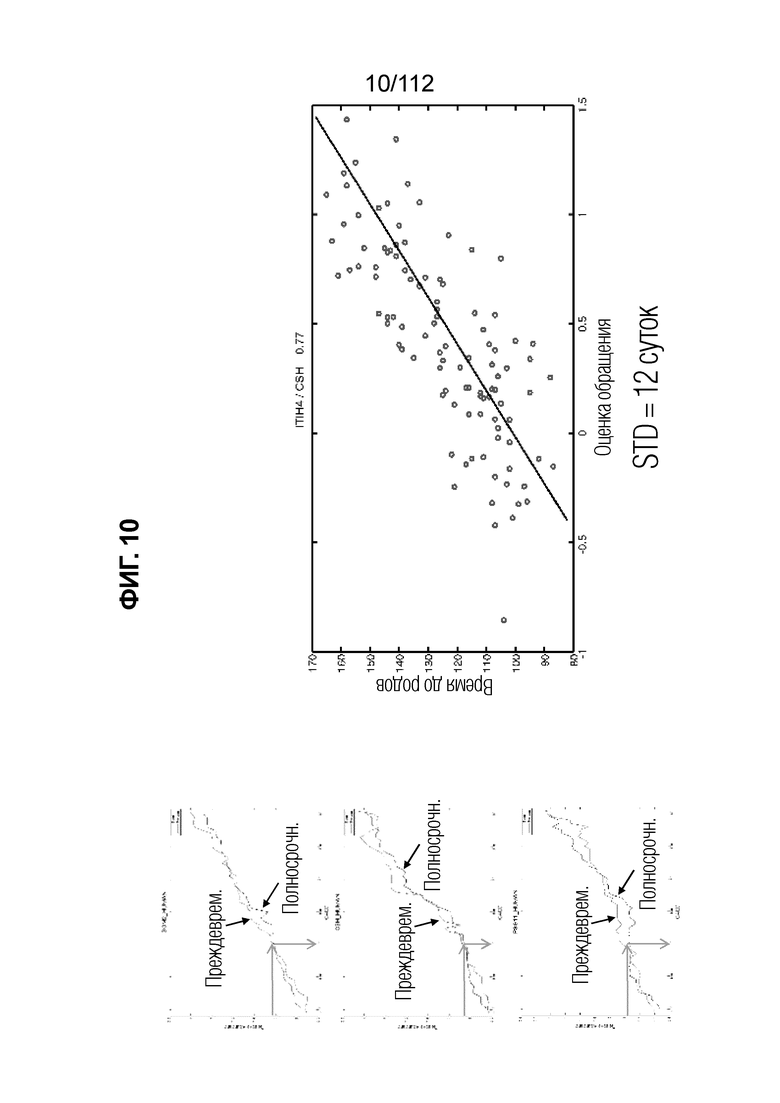
[0054] Фиг. 10. Хронология беременности и время до родов. Множество анализируемых веществ, которые повышаются при беременности, но не различаются в случаях PTB и контролях, можно использовать для того, чтобы датировать беременность биохимически. Биохимическое датирование может быть полезным для подтверждения датирования по дате последнего менструального цикла или ультразвукового датирования или перед последующим определением риска sPTB, предсказания TTB или GAB.
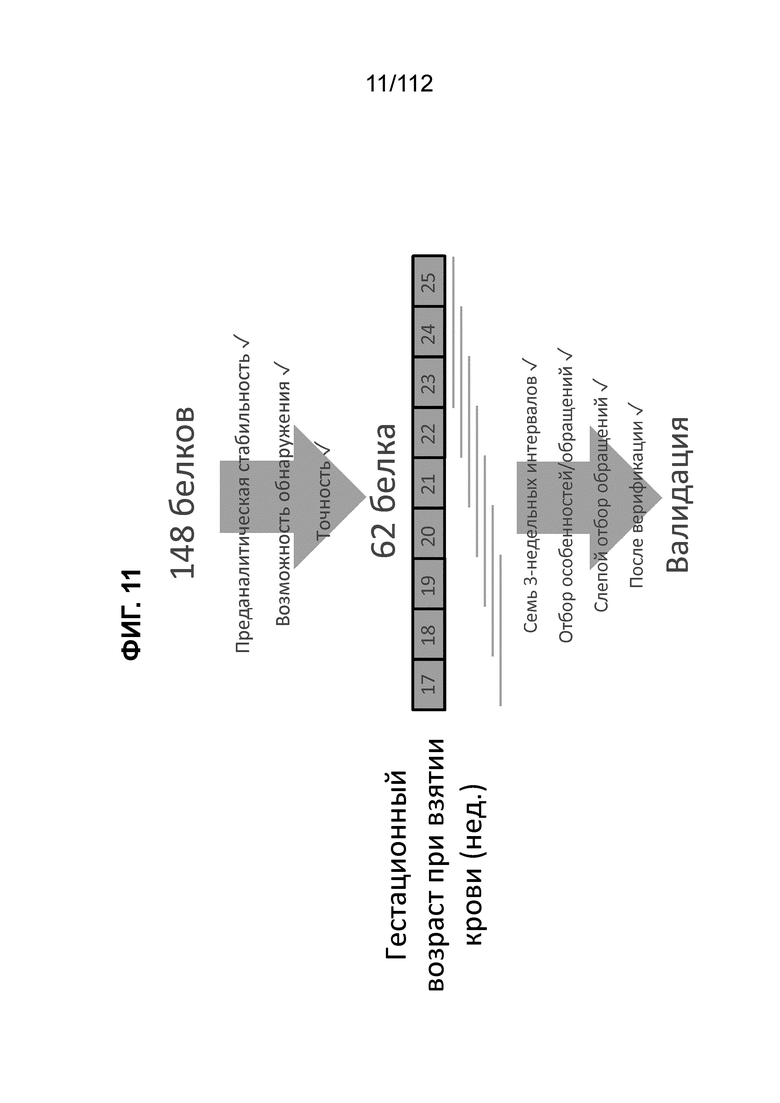
[0055] Фиг. 11. Разработка классификатора. На фиг. 11 представлены критерии для разработки классификаторов.
[0056] Фиг. 12. Покрытие пути в исследовательском анализе. На фиг. 12 представлено распределение белков по пути.
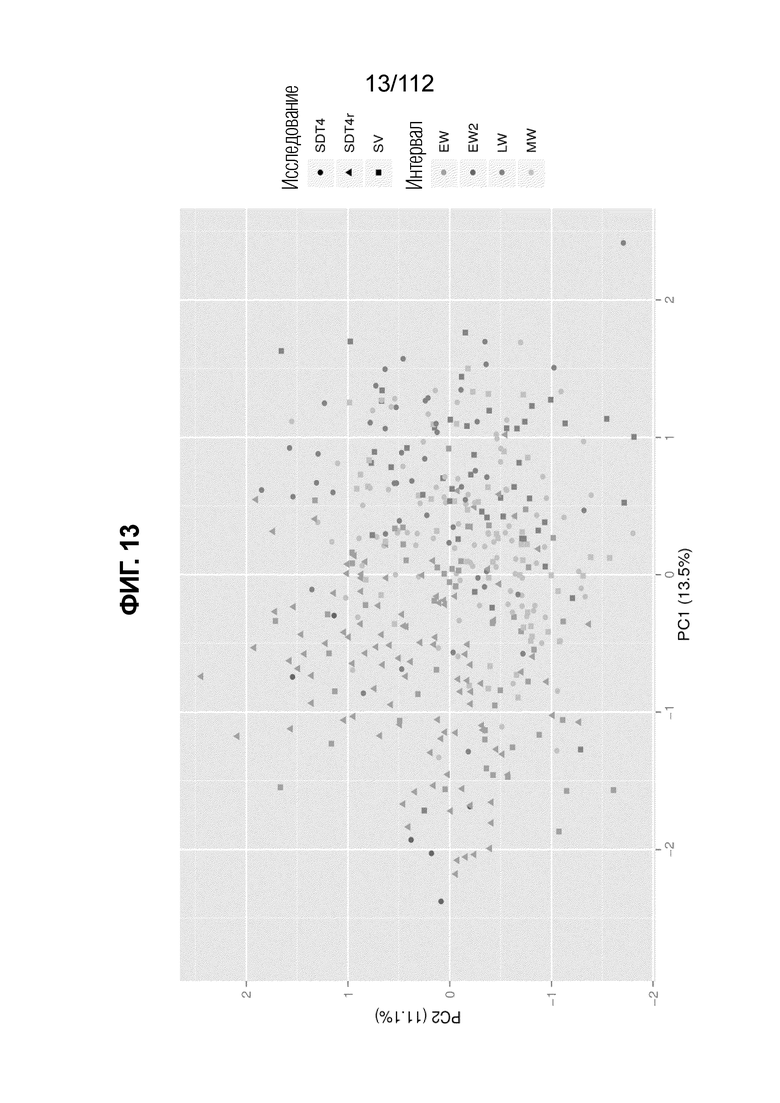
[0057] Фиг. 13. PCA для данных исследования обнаруживает изменения в интервалах взятия крови и, следовательно, указывает на то, что высоко мультиплексный анализ чувствителен к гестационному возрасту.
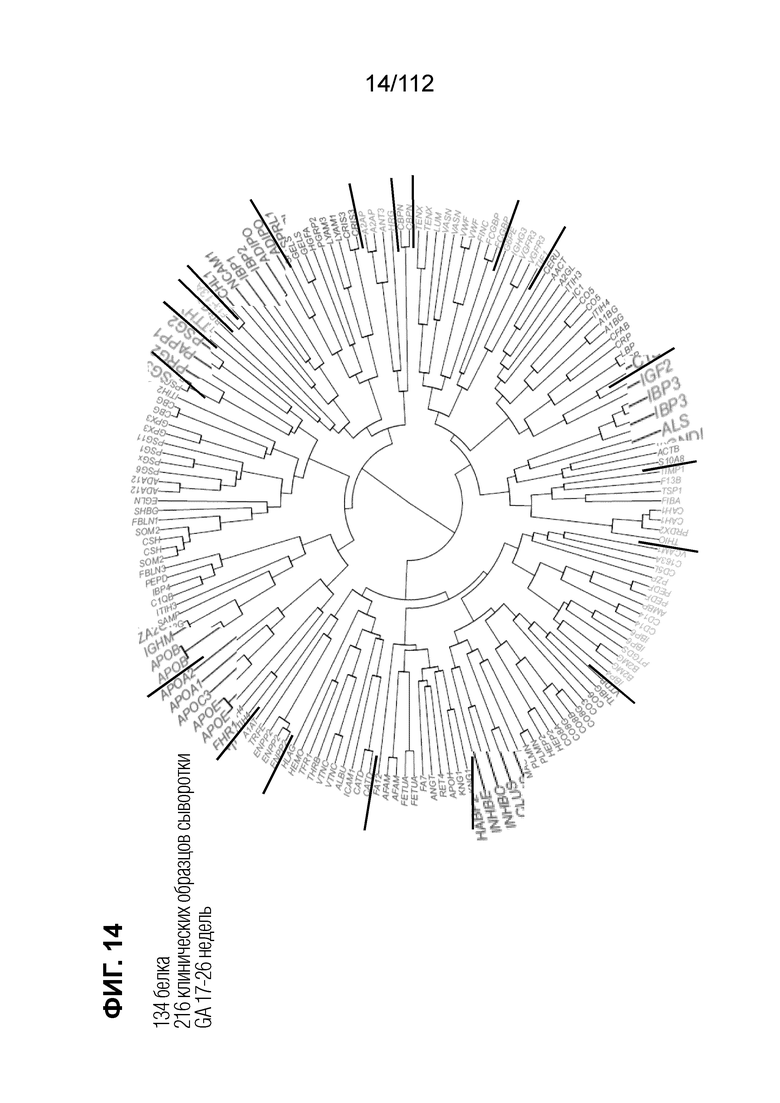
[0058] Фиг. 14. Иерархическая кластеризация белков, измеряемых в образцах исследования.
[0059] Фиг. 15. Ветвь плацентарных белков в более крупном кластере. Справа перечислен блок генов, экспрессируемых во время беременности, которые идентифицировал Thompson, а слева показано, что исследовательский протеомный анализ сыворотки воспроизводит скоррелированную экспрессию этого блока. (Thompson et al., Genome Res. 12(10):1517-1522 (2002).
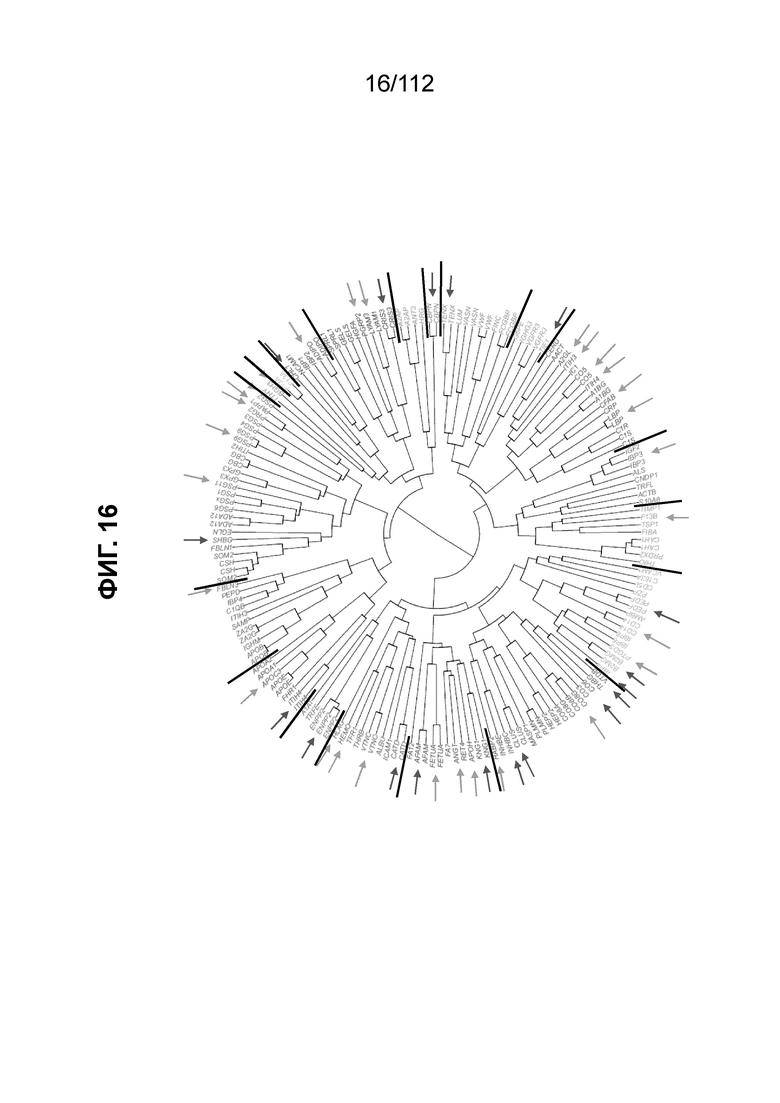
[0060] Фиг. 16. Образцы белков PreTRMTM с нарушенной регуляцией.
[0061] Фиг. 17. Основные аспекты биологии связывающего половые гормоны глобулина (SHGB). SHBG экспрессирован в плацентарных клетках (справа). SHBG может отвечать за контроль уровней свободного тестостерона и уровней эстрогена в фето-плацентарном компартменте (слева).
[0062] Фиг. 18. Взаимодействия IBP4, IGF2, PAPP-A и PRG2. IBP4 представляет собой отрицательный регулятор для IGF2. IGF2 высвобождает IBP4 посредством протеолиза, опосредованного PAPPA. Низкие уровни PAPPA вовлечены в IUGR и PE. Повышенные уровни IBP4 отражают супрессированную активность IGF2. В случаях PTB имеют место супрессированные уровни PAPPA, PRG2 и повышенные уровни IBP4.
[0063] Фиг. 19. Связывающий инсулиноподобный фактор роста белок 4 (IBP4). IBP4 находится под повышающей регуляцией в случаях PTB. IGF2 стимулирует пролиферацию, дифференцировку и инвазию EVT на ранней беременности. Активность IGF важна для нормального образования плаценты и роста плода. IBP4 опосредует аутокринный и паракринный контроль активности IGF2 на трансплацентраном барьере. Активность IGF2, экспрессируемого цитотрофобластами, уравновешивается с помощью IBP, продуцируемого децидуальными клетками. Повышенный IBP4 и пониженный в IGF2 в 1-м триместре коррелированны с нарушением плацентарной функции (например, IUGR/SGA).
[0064] Фиг. 20. Корреляция MS и ELISA для IBP4, SHBG и CHL1. Масс-спектрометрия и ELISA находятся в хорошем соответствии по ключевым анализируемым веществам. Соответствие двух независимых платформ говорит о надежности измерения анализируемых веществ.
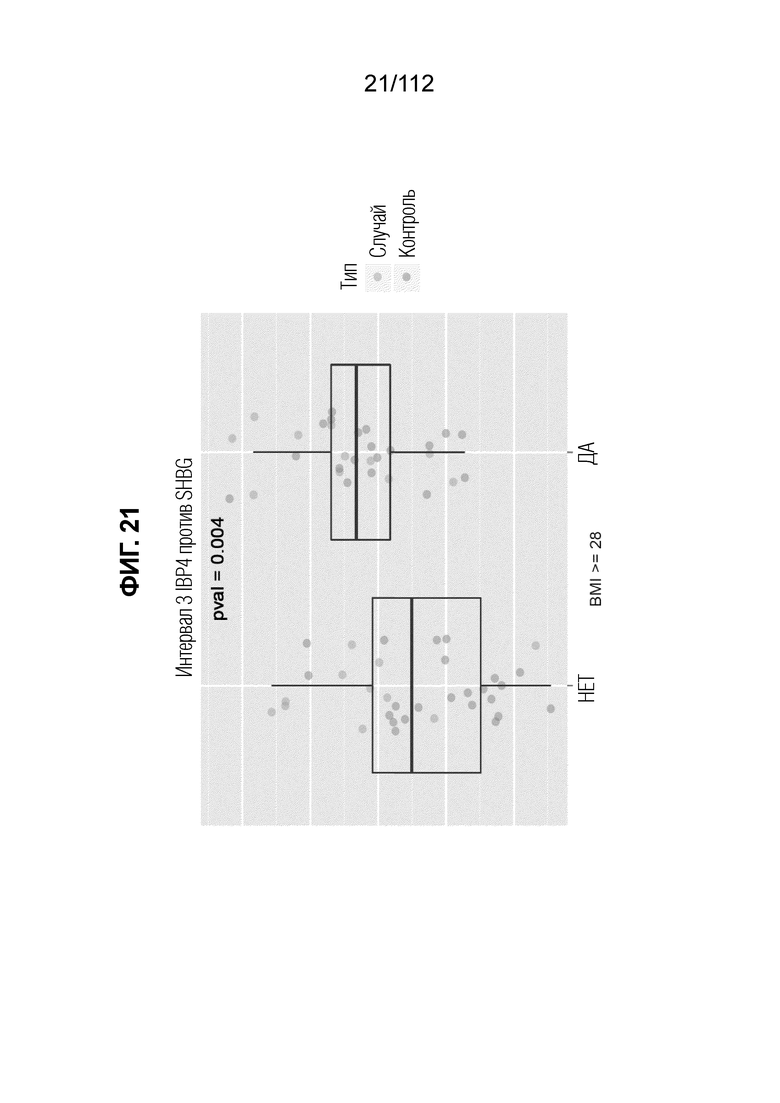
[0065] Фиг. 21. Классификация PTB по IBP4/SHBG в образцах исследования из 19-21 недель GABD. Образцы исследования для недель 19-21 беременности делили по высокому и низкому BMI. Значения обращения IBP4/SHBG выше в категории высокого BMI из-за более низких значений SHBG. Разделение случаев и контролей больше при более низком BMI.
[0066] Фиг. 22. Супрессированные уровни SHBG в случаях PTB при низком BMI. Линейные приближения уровней SHBG в сыворотке у PAPR-субъектов в зависимости от GABD. Уровни SHBG супрессированы при высоком BMI. Уровни SHBG растут на протяжении беременности. Случаи PTB при низком BMI имеют сниженные уровни SHBG, которые растут на протяжении беременности с повышенной скоростью.
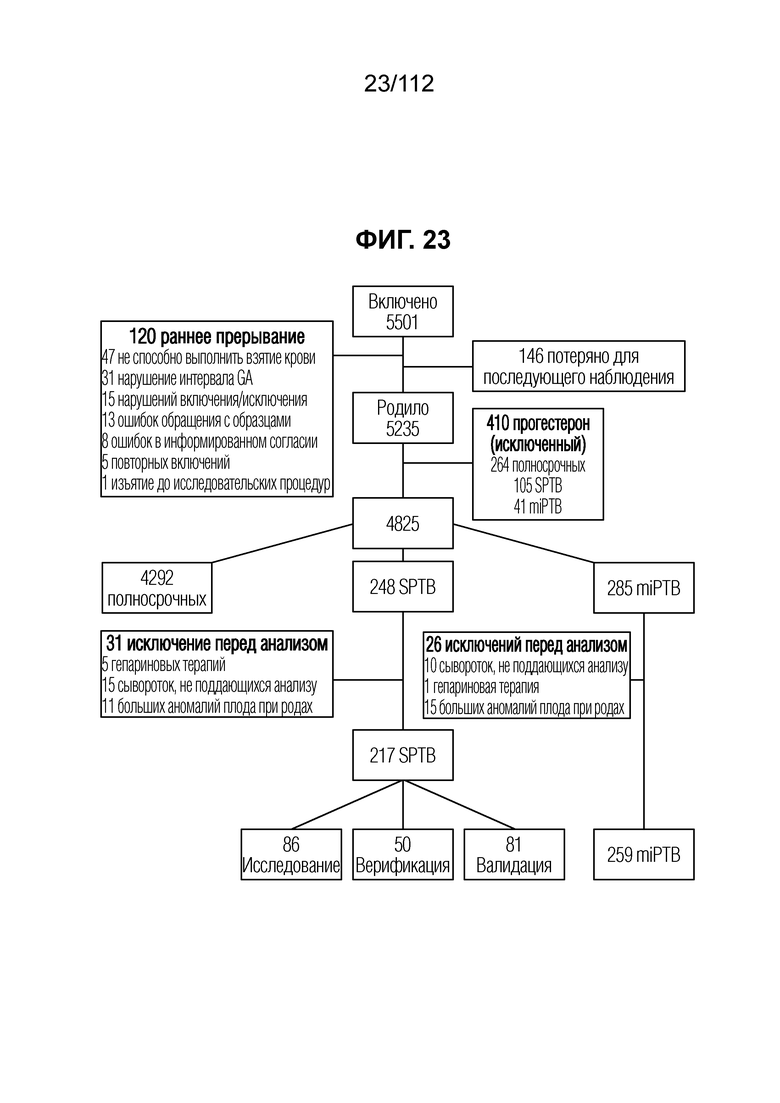
[0067] На фиг. 23 краткое изложено распределение исследуемых субъектов при PAPR.
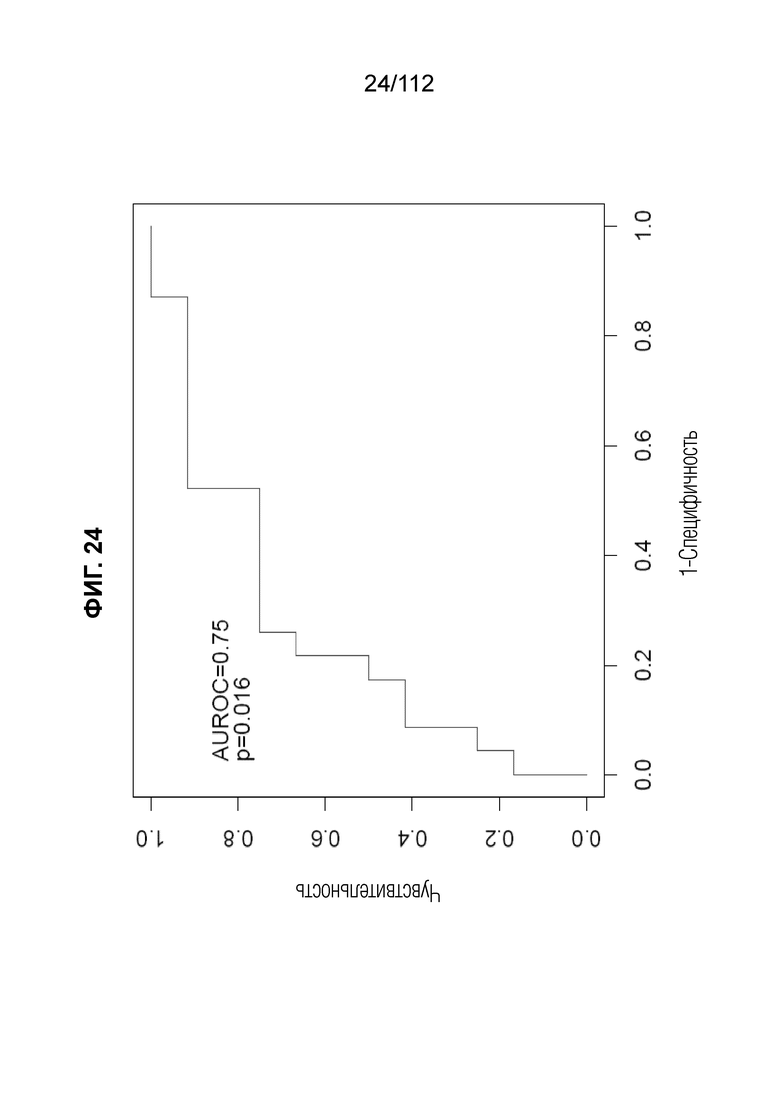
[0068] На фиг. 24 представлена ROC-кривая и соответствующее значение AUC с использованием предиктора IBP4/SHBG для того, чтобы классифицировать BMI-стратифицированный валидационный набор образцов.
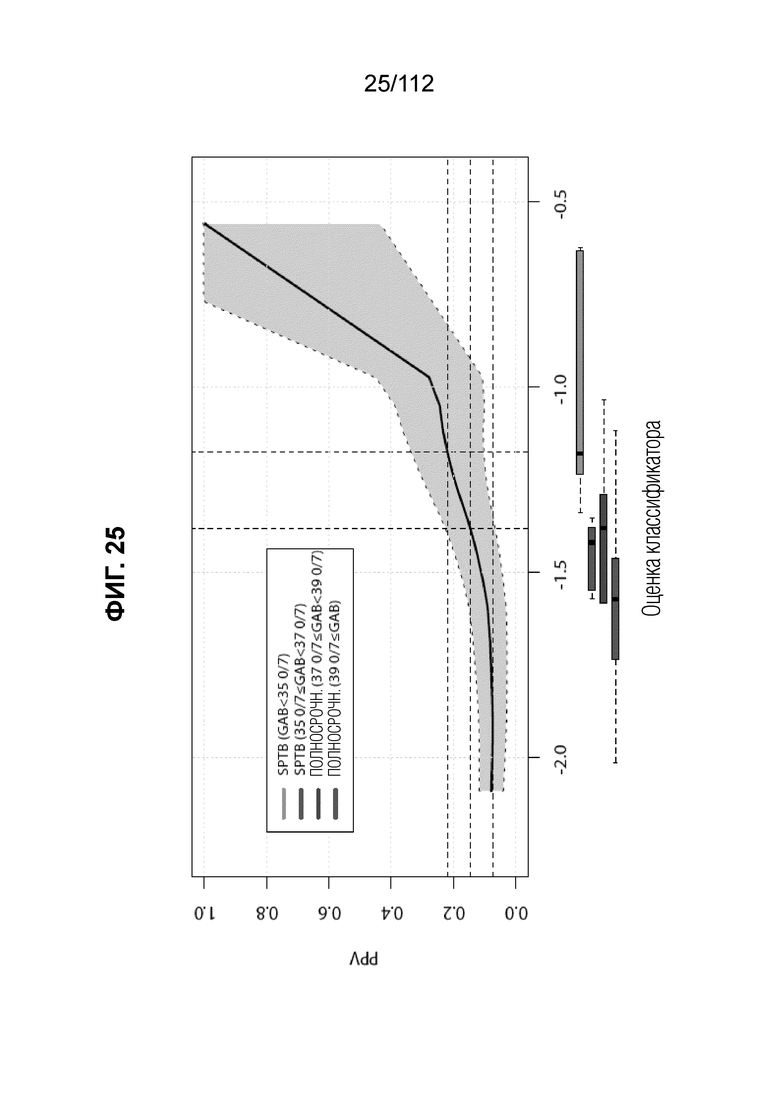
[0069] На фиг. 25 представлено скорректированное по болезненности положительное прогностическое значение (PPV), мера клинического риска, как функция оценки предиктора. Вычисленная связь оценки предиктора и PPV позволяет определять вероятность риска sPTB для какого-либо неизвестного субъекта. Верхняя (пурпурная) линия под графиком кривой риска соответствует GAB <35 0/7 недель; вторая линия (красная) сверху соответствует GAB между 35 0/7 и 37 0/7/ неделями; третья линия (зелена) с соответствует GAB между 37 0/7 и 39 0/7/ неделями; четвертая линия (синяя) сверху соответствует GAB 39 0/7 недель≤GAB.
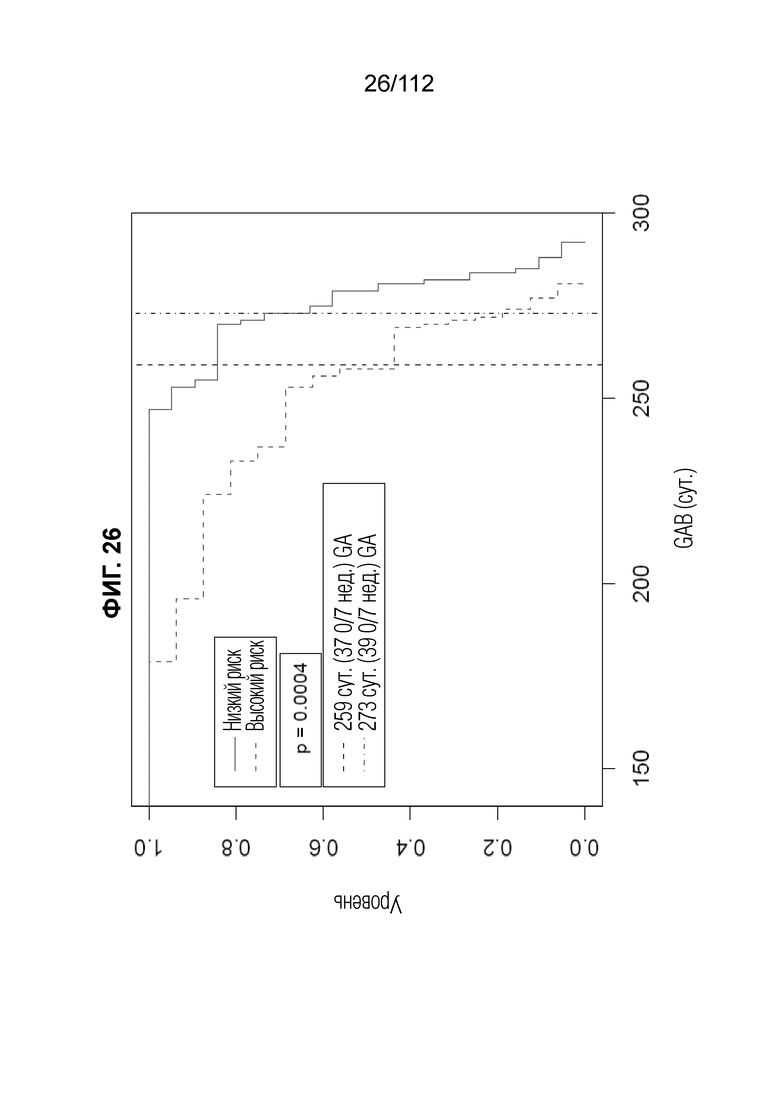
[0070] На фиг. 26 представлена частота родов для групп высокого и низкого риска в качестве событий в анализе Каплана-Мейера. Высокий и низкий риск определяли как выше или ниже относительного риска как 2× риска sPTB в усредненной популяции (=14,6%) из данных на фиг. 25.
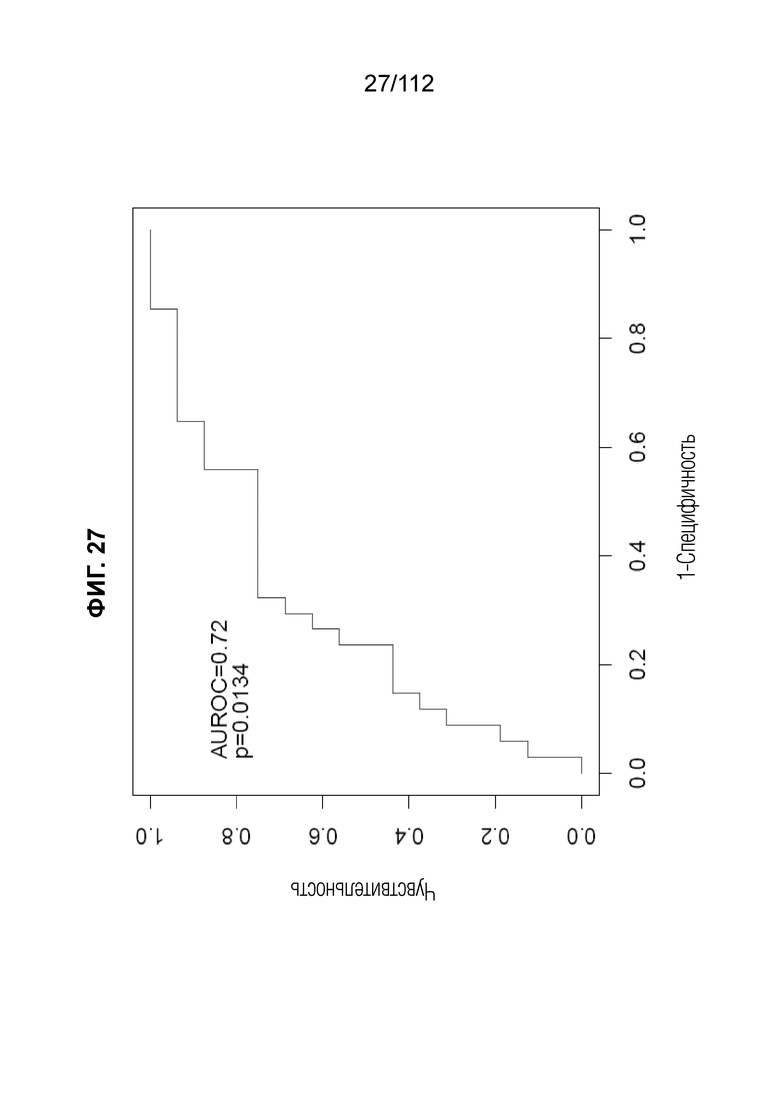
[0071] На фиг. 27 представлена ROC-кривая, соответствующая эффективности предиктора при использовании комбинации субъектов из слепого верификационного и валидационного анализа в пределах оптимального интервала BMI и GA. ROC-кривая для комбинированного образца соответствует AUROC, равной 0,72 (p=0,013).
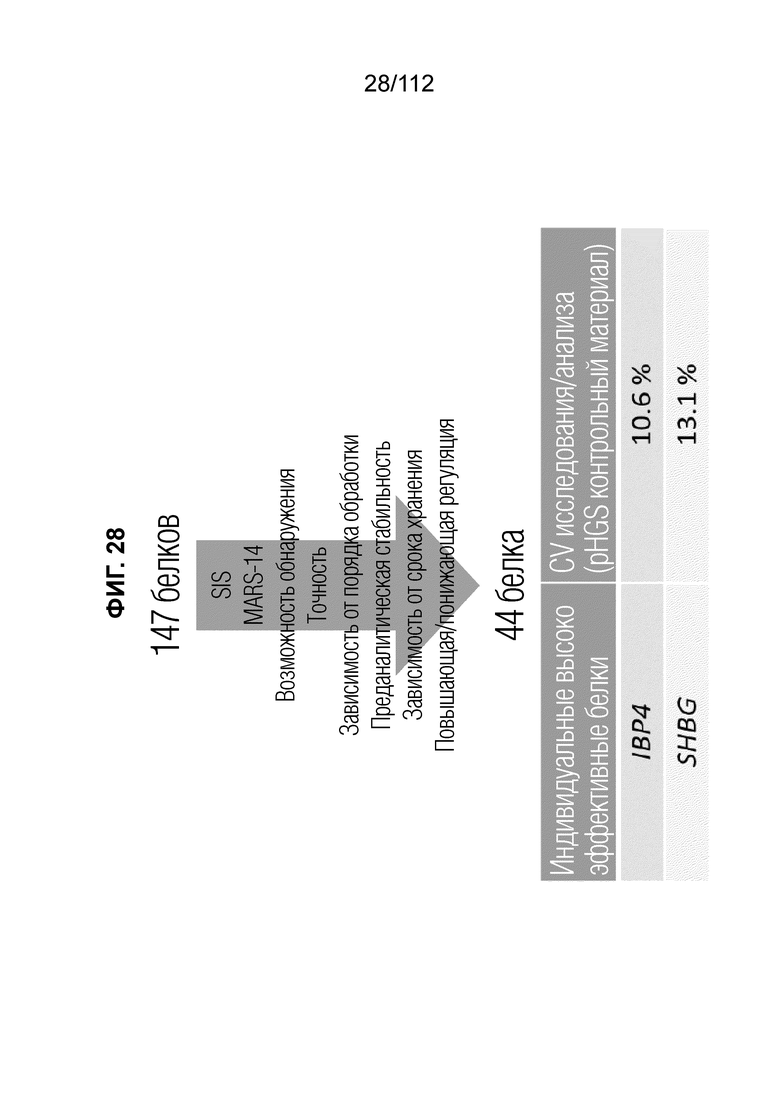
[0072] На фиг. 28 представлены 44 белка, которые находились под повышающей или понижающей регуляцией в перекрывающихся 3-недельных интервалах GA и проходили аналитические фильтры.
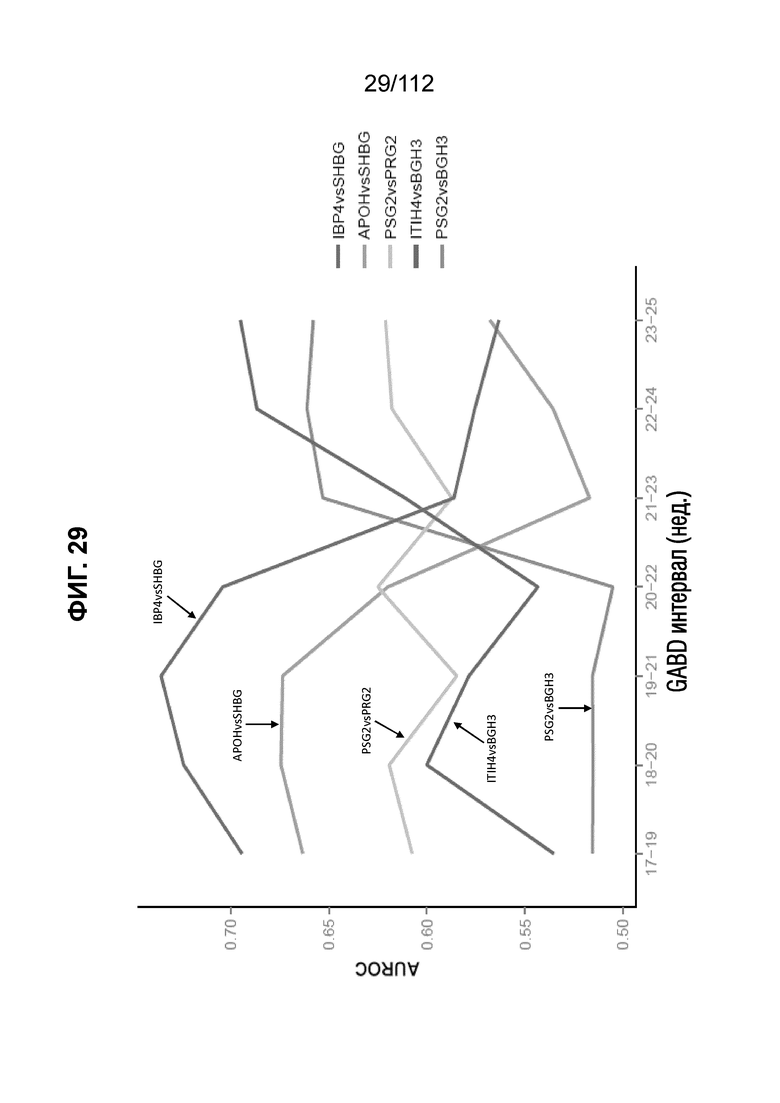
[0073] На фиг. 29 представлено наиболее эффективное обращение из всех, IBP4/SHBG, которое имело AUROC=0,74 в интервале от 19 0/7 до 21 6/7.
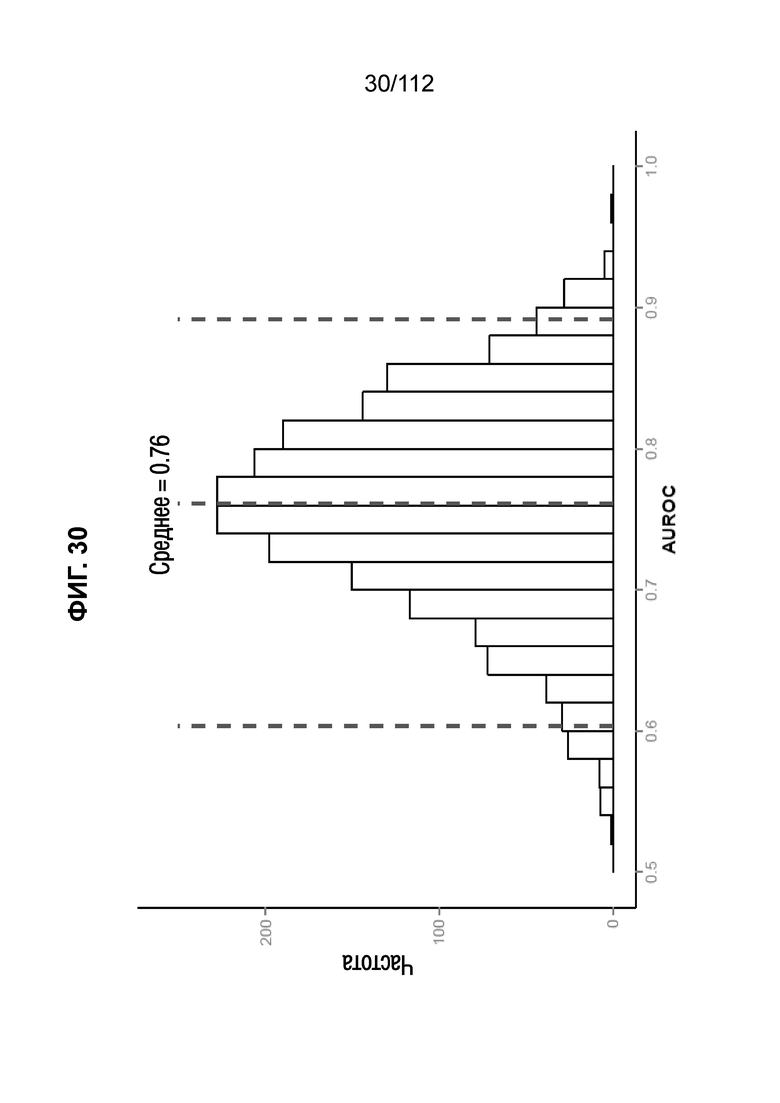
[0074] На фиг. 30 представлена средняя AUROC, равная 0,76, которую получали после 2000 итераций бутстрэпа. Слепая IBP4/SHBG AUROC эффективность на верификационных образцах составляла 0,77 и 0,79 для всех субъектов и BMI-стратифицированных субъектов, соответственно, в хорошем соответствии с эффективностью, полученной в исследовании. После слепой верификации, образцы исследования и верификационные образцы комбинировали для определения эффективности бутстрэпа.
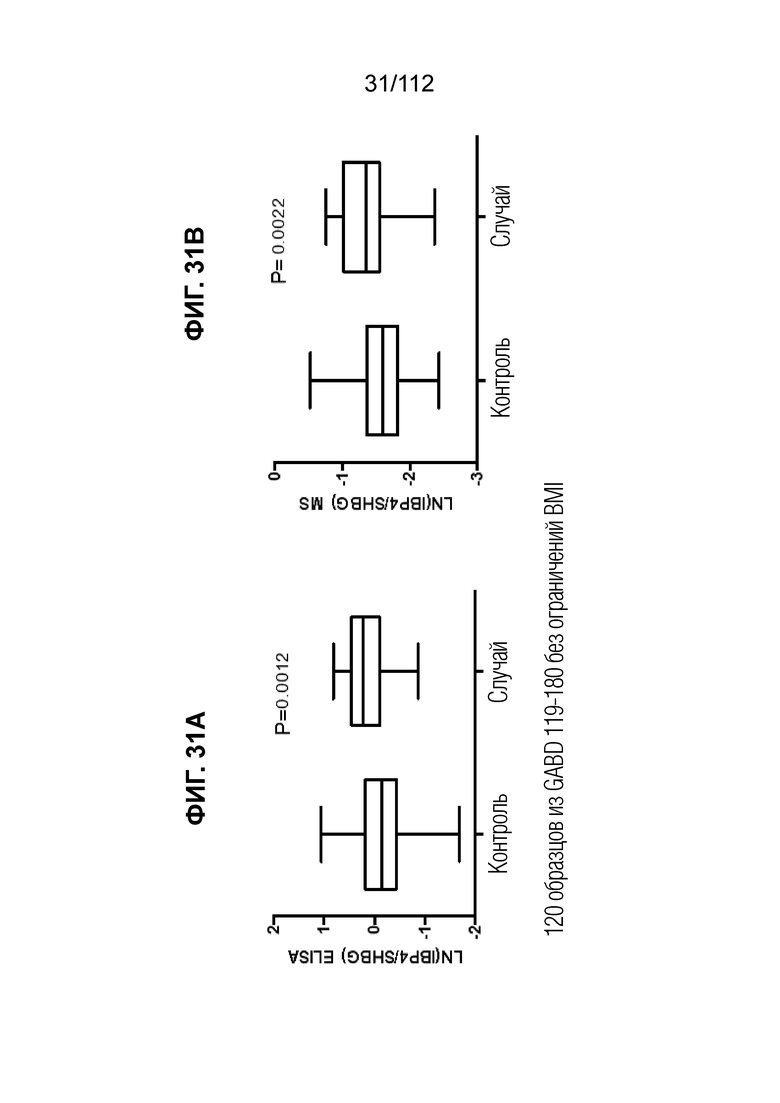
[0075] На фиг. 31 представлено разделение случаев sPTB и контролей, полученное по значениям оценок MS в сравнении с ELISA
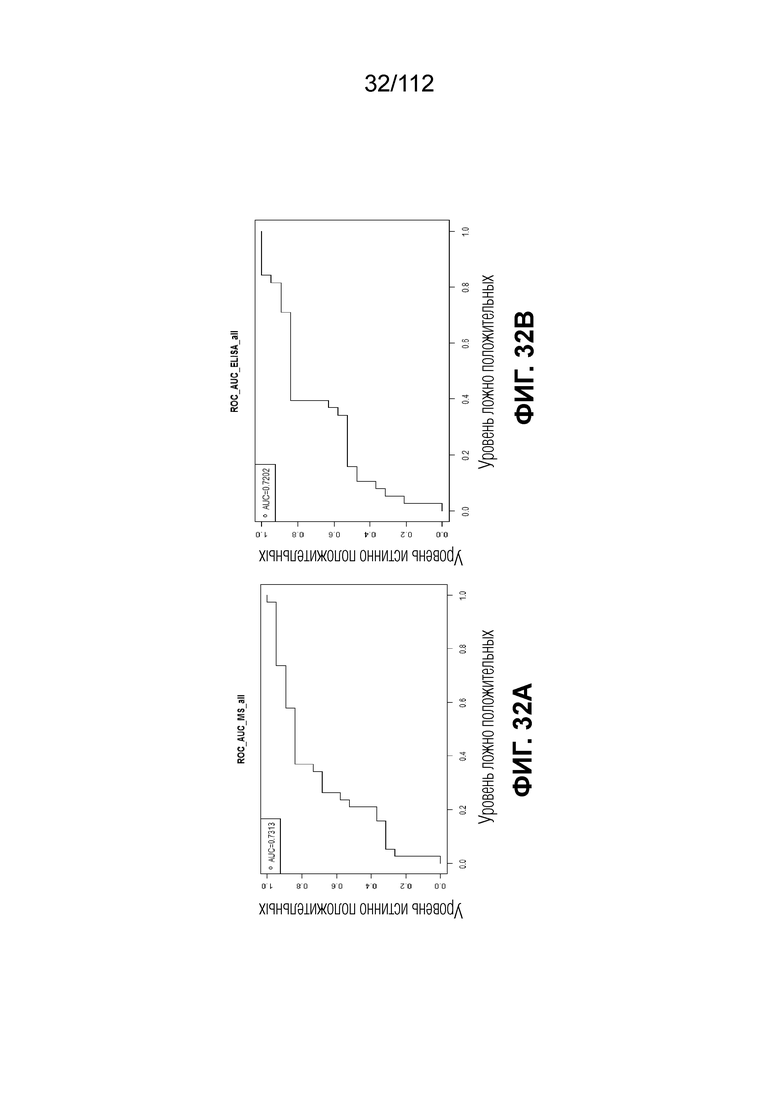
[0076] На фиг. 32 представлен иммунологический анализ в сравнении с MS ROC анализами без ограничения по BMI.
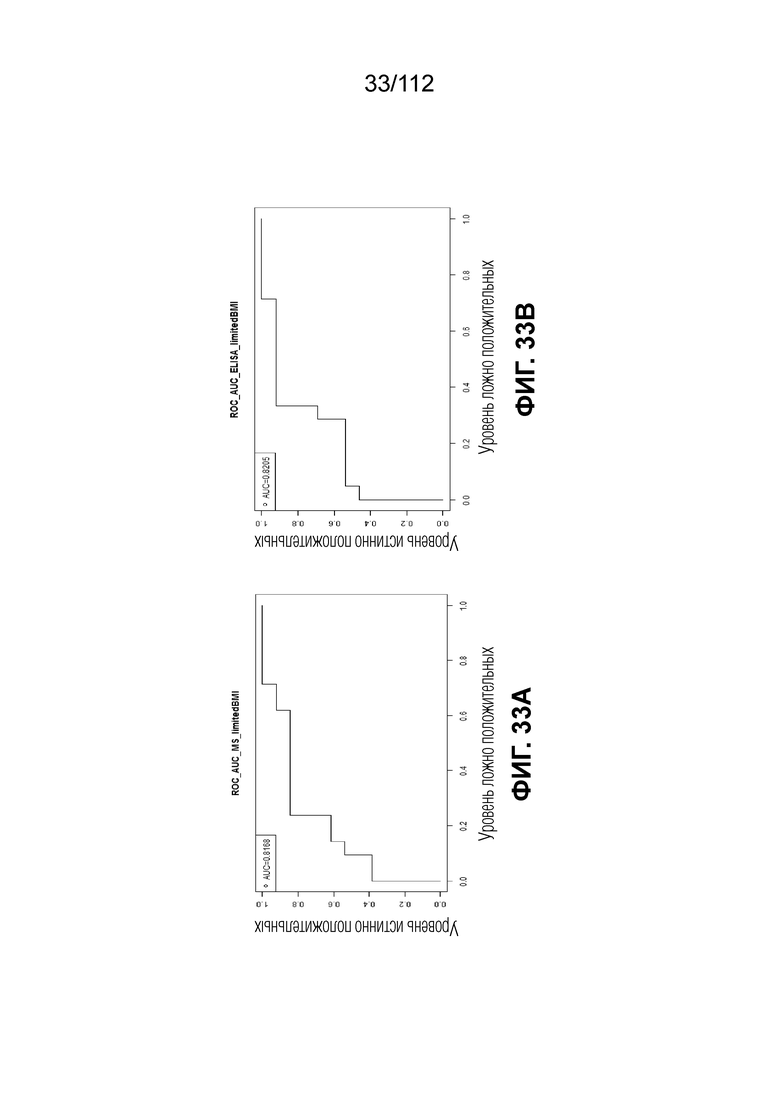
[0077] На фиг. 33 представлен иммунологический анализ в сравнении с MS ROC анализами для BMI выше 22 и меньше или равных 37.
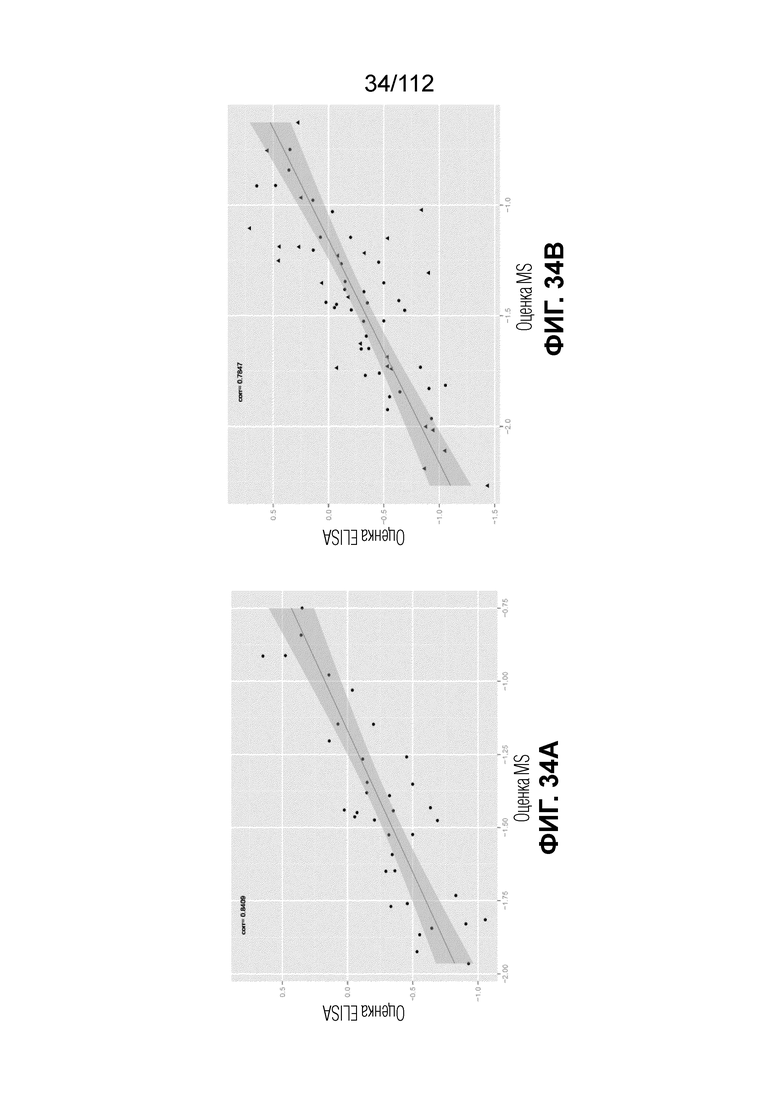
[0078] На фиг. 34 представлена корреляция между значениями оценок MS и ELISA, полученными для IBP4/SHBG, в пределах GABD 133-146, для BMI-стратифицированных субъектов (слева) и всех субъектов (справа).
[0079] На фиг. 35 представлено разделение контролей и случаев (BMI-стратифицированных) по ELISA и MS.
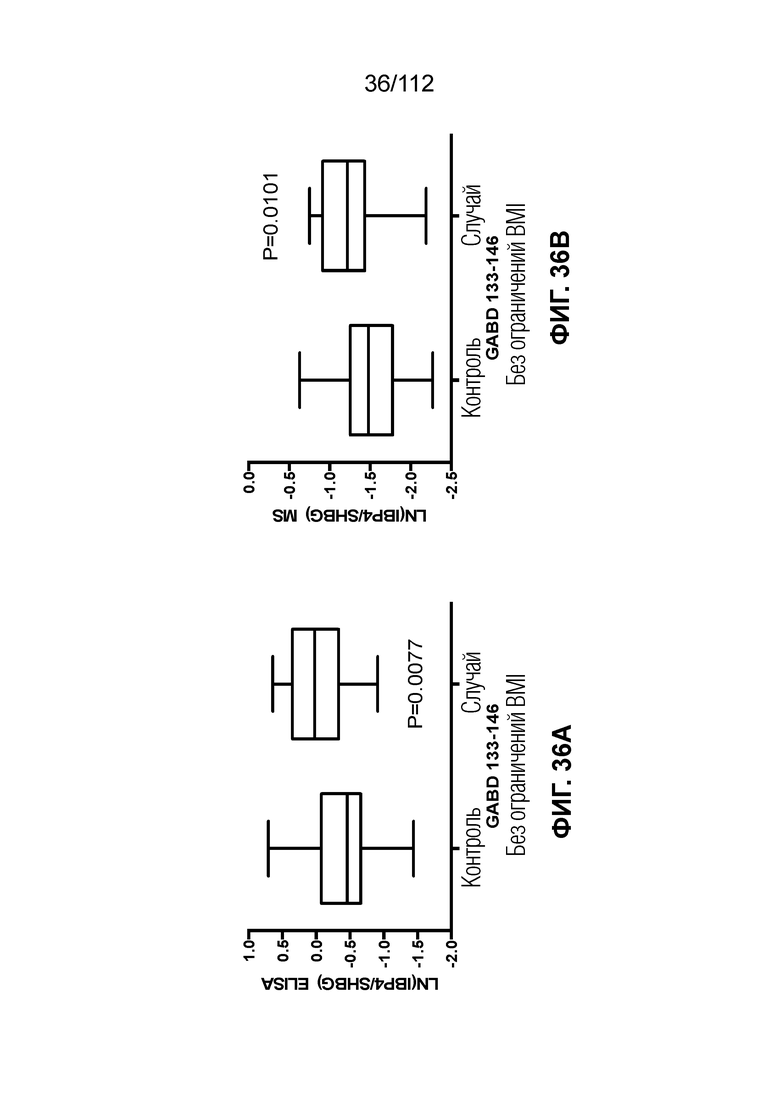
[0080] На фиг. 36 представлено разделение контролей и случаев (все BMI) по ELISA и MS
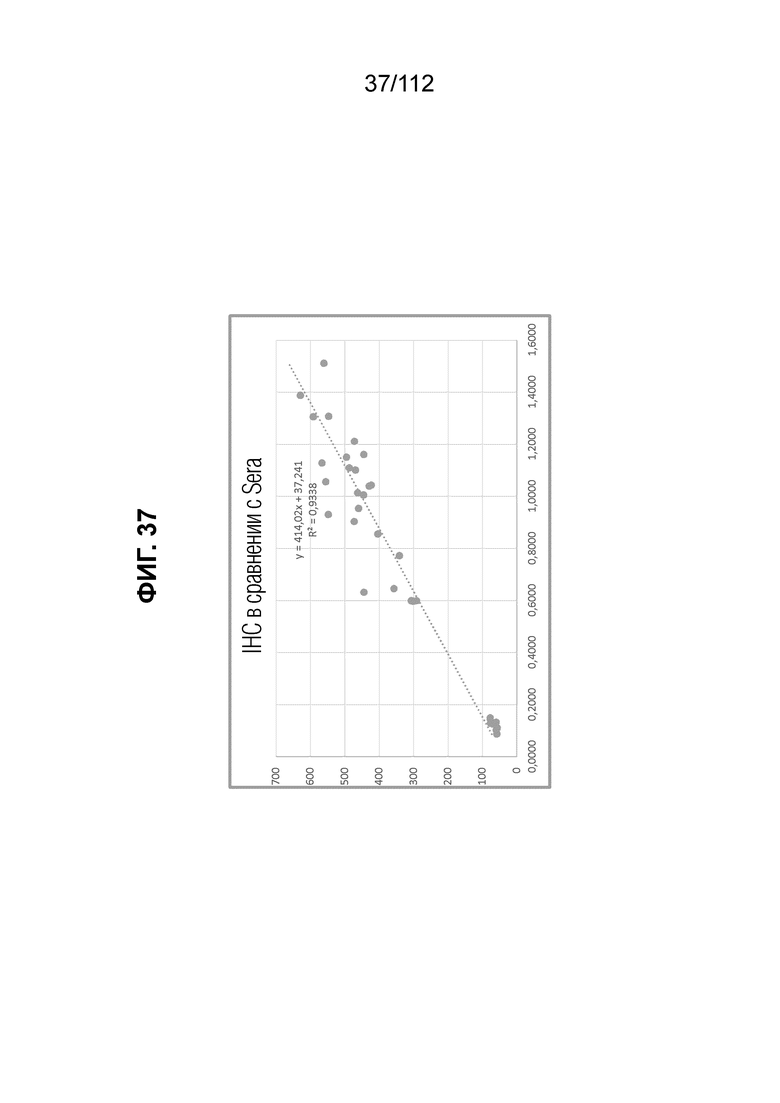
[0081] На фиг. 37 представлено сравнение измерений SHBG с помощью Abbott Architect CMIA, полуавтоматизированных приборов иммунологического анализа и способа протеомного анализа Sera Prognostics с использованием иммунного истощения образцов, ферментативного расщепления и анализа на Agilent 6490 Mass Spectrometer.
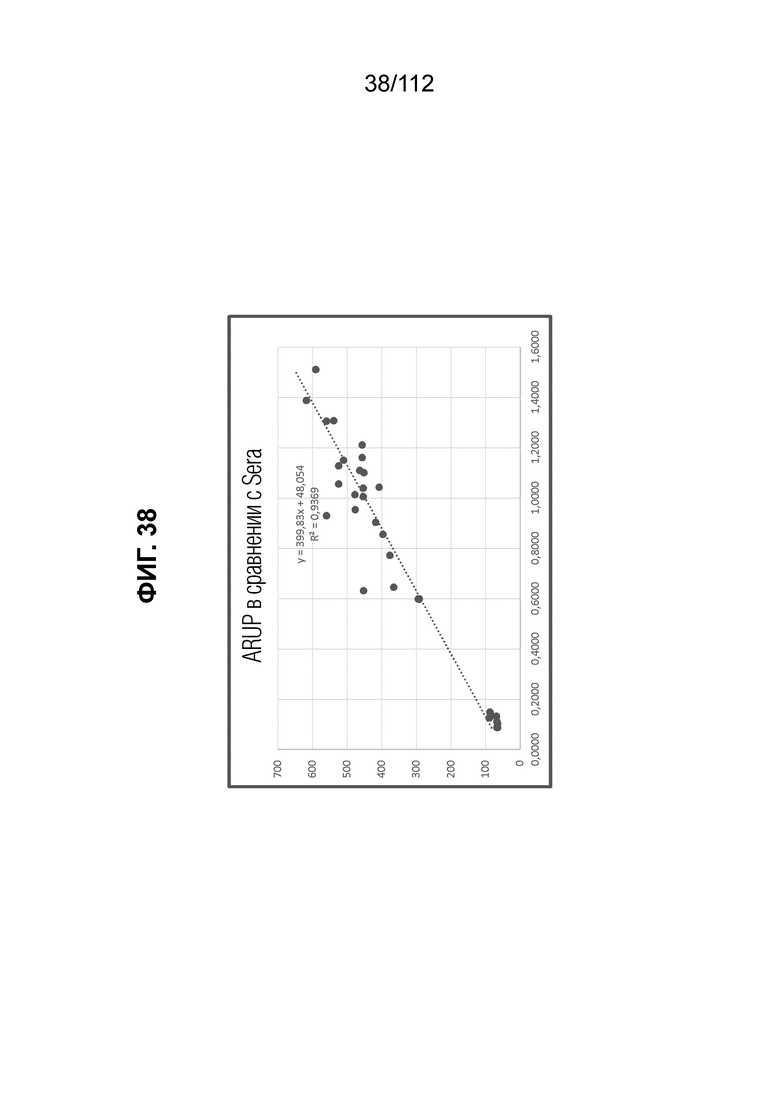
[0082] На фиг. 38 представлено сравнение измерений SHBG с помощью анализатора Roche cobas e602, полуавтоматизированных приборов иммунологического анализа и способа протеомного анализа Sera Prognostics с использованием иммунного истощения образцов, ферментативного расщепления и анализа на Agilent 6490 Mass Spectrometer.
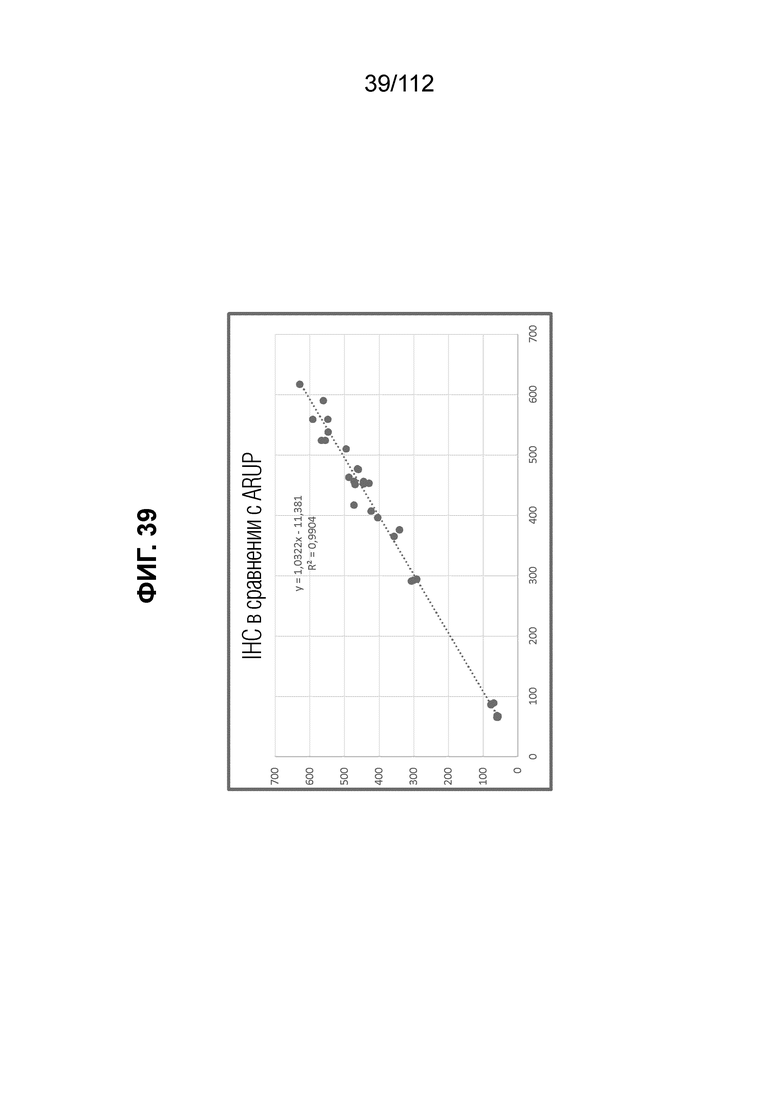
[0083] На фиг. 39 представлено сравнение измерений SHBG с помощью Abbott Architect CMIA и анализатора Roche cobas e602*, оба полуавтоматизированные приборы иммунологического анализа.
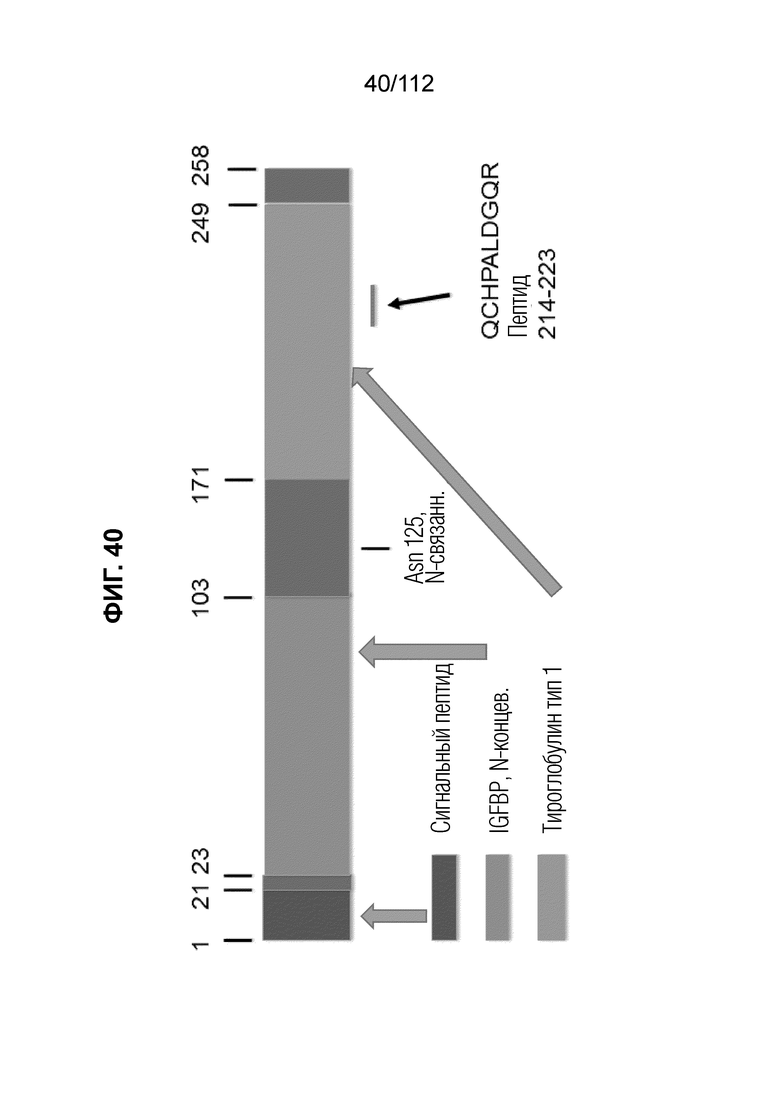
[0084] На фиг. 40 представлены доменные и структурные характеристики самой длинной изоформы белка IBP4 (Uniprot: P22692). Пептид IBP4 QCHPALDGQR (а. о. 214-223) (SEQ ID № 2) расположен внутри домена тироглобулина 1 типа. IBP4 имеет один участок N-связанного гликозилирования в остатке 125.
[0085] На фиг. 41 отмечено положение пептида QCHPALDGQR (SEQ ID № 2) в двух изоформах IBP4 (SEQ ID №№ 158 и 159, соответственно, в порядке появления).
[0086] На фиг. 42 представлены доменные и структурные характеристики самой длинной изоформы белка SHBG (Uniprot: P04278). Пептид SHBG IALGGLLFPASNLR (а. о. 170-183) (SEQ ID № 18) расположен в первом ламин G-подобном домене. SHBG имеет три участка гликозилирования; два N-связанных участка в остатках 380 и 396; один O-связанный участок в остатке 36.
[0087] На фиг. 43 отмечено положение пептида IALGGLLFPASNLR (SEQ ID № 18) в экзоне 4 в семи изоформах SHBG (SEQ ID №№ 160, 160, 160, 160, 160, 160 и 161, соответственно, в порядке появления).
[0088] На фиг. 44 представлен усредненный коэффициент ответа для уровней IBP4 отдельно для случаев sPTB и полносрочных контролей в зависимости от гестационного возраста при взятии крови (GABD). Перекрестные данные исследования анализировали посредством сглаживания с использованием скользящего 10-суточного окна. Сигнал случая в сравнении с контролем соответствует приблизительной максимальной 10% разнице.
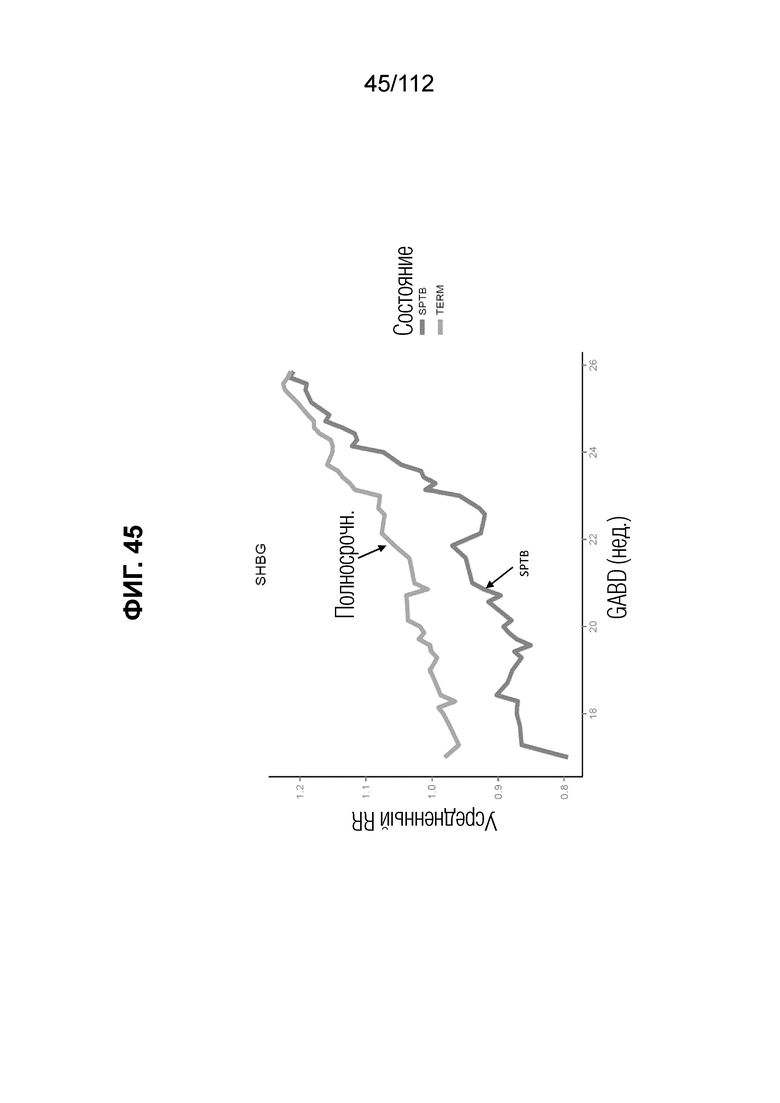
[0089] На фиг. 45 представлен усредненный коэффициент ответа для уровней SHBG отдельно для случаев sPTB и полносрочных контролей в зависимости от гестационного возраста при взятии крови (GABD). Перекрестные данные исследования анализировали посредством сглаживания с использованием скользящего 10-суточного окна. Сигнал случая в сравнении с контролем соответствует приблизительной максимальной 10% разнице.
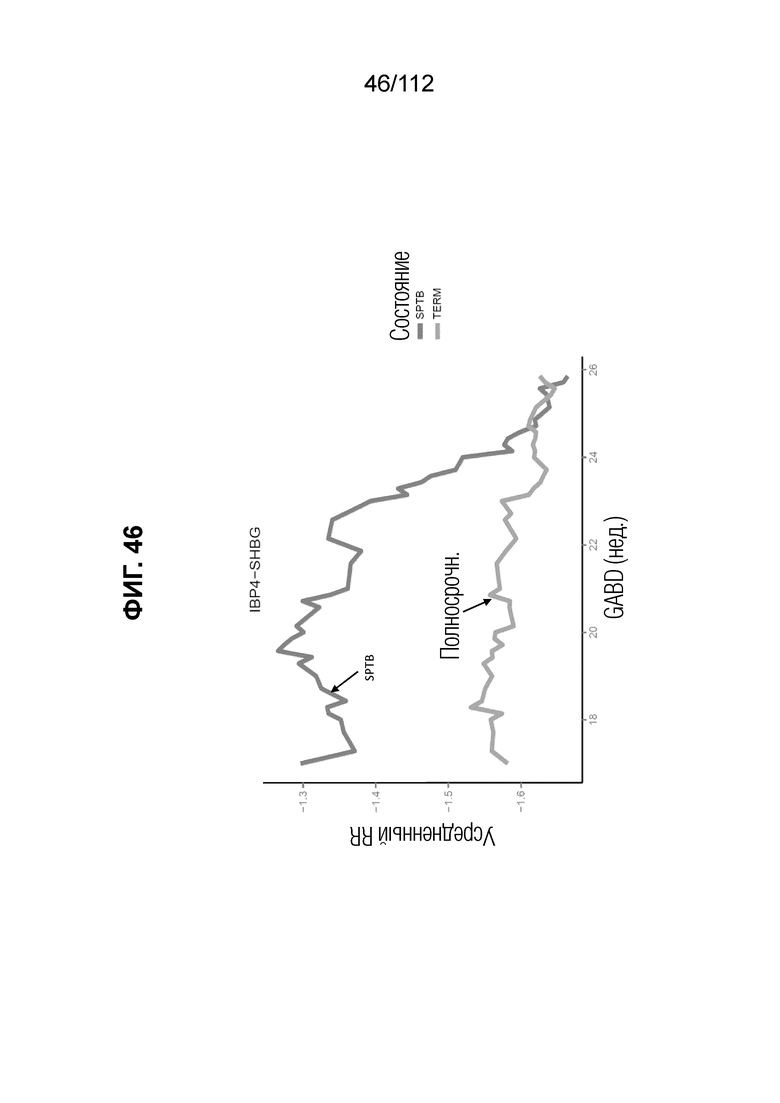
[0090] На фиг. 46 представлена оценка предиктора IBP4/SHBG отдельно для случаев sPTB и полносрочных контролей в зависимости от гестационного возраста при взятии крови (GABD). Перекрестные данные исследования анализировали посредством сглаживания с использованием скользящего 10-суточного окна. Максимальная разница между двумя кривым соответствует приблизительно 20% разнице, по сравнению с приблизительной 10% разницей в сигнале для индивидуальных анализируемых веществ (фиг. 45 и 46). Эти данные демонстрируют усиление диагностического сигнала, получаемого с использованием стратегии обращения IBP4/SHBG.
[0091] На фиг. 47 представлено усиление диагностического сигнала в результат формирования многих различных обращений. Чтобы в целом исследовать, усиливает ли формирование обращений диагностический сигнал, авторы изобретения изучали диагностическую эффективность обращений, формируемых многими различными белками посредством ROC анализа. Сверху показан диапазон значений AUC (случай sPTB в сравнении с полносрочным контролем) с использованием массивов данных от образцов, собранных между 19/0 неделями и 21/6 неделей беременности. Смежные диаграммы размаха демонстрируют диапазон ROC эффективности для индивидуальных белков под повышающей регуляцией и понижающей регуляцией, используемых для того, чтобы формировать связанные обращения. Аналогичным образом, снизу представлены p-значения, полученные для критерия Уилкоксона (случай sPTB в сравнении с полносрочными контролями) для обращений, которые более значимы, чем таковые для соответствующих индивидуальных белков.
[0092] На фиг. 48 представлен аналитический коэффициент вариации (CV) для меры индивидуальных коэффициентов ответа IBP4 и SHBG и для вычисленной соответствующей оценки обращения. Объединенные образцы сывороток контролей от беременных доноров (pHGS), лишенные биологической вариабельности, анализировали в нескольких партиях и для нескольких суток. Вариабельность обращения меньше вариабельности, связанной с индивидуальными белками. Эти данные отражают это формирование контролей обращения для аналитической вариабельности, которая возникает во время лабораторной обработки образцов. Аналитическая вариабельность не является биологическим феноменом.
[0093] На фиг. 49 представлены аналитические CV для нескольких обращений и их индивидуальные белки под повышающей и понижающей регуляцией. Чтобы в целом исследовать, усиливает ли формирование обращений диагностический сигнал, авторы изобретения изучали ROC эффективность (AUC) высоко эффективных обращений (AUC >0,6), сформированных с помощью соотношений нескольких белков. Сверху показан диапазон значений AUC (случай sPTB в сравнении с полносрочным контролем) с использованием массивов данных от образцов, собранных между 19/0 неделями и 21/6 неделей беременности. Смежные диаграммы размаха показывают диапазон ROC эффективности для индивидуальных белков под повышающей регуляцией и понижающей регуляцией, которые использовали для того, чтобы формировать связанные обращения. Аналогичным образом, p-значения, полученные для критерия Уилкоксона (случай sPTB в сравнении с полносрочными контролями) для обращений, более значимы, чем таковые для соответствующих индивидуальных белков.
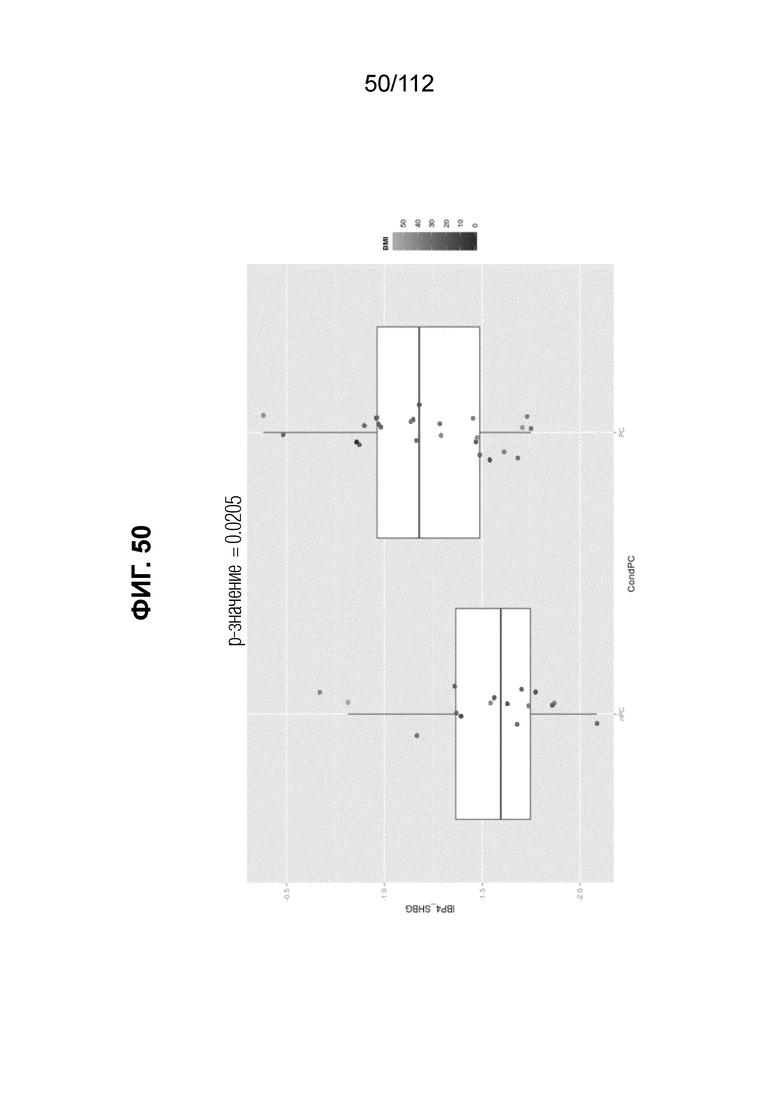
[0094] На фиг. 50 представлено сравнение оценки PreTRMTM для субъектов, аннотированных как имеющих медицинские показания для предэклампсии, в сравнении с другими показаниями.
[0095] На фиг. 51 представлена таблица метрики эффективности предиктора IBP4/SHBG в валидационном наборе образцов (BMI>22≤37). Используя различные границы для определения случаев (ниже порога) и контролей (выше порога), определяли чувствительность, специфичность предиктора, площадь под ROC-кривой (AUC) и отношение шансов.
[0096] На фиг. 52 представлена тепловая карта интенсивности обращений с аннотацией для диабета. Красные стрелки показывают случаи диабета. Образцы перечислены внизу, случаи PTB справа и полносрочные роды слева экрана. Пациенты с диабетом кластеризованы справа, что указывает на возможность построения диагностического теста из биологических маркеров для предсказания диабета беременных.
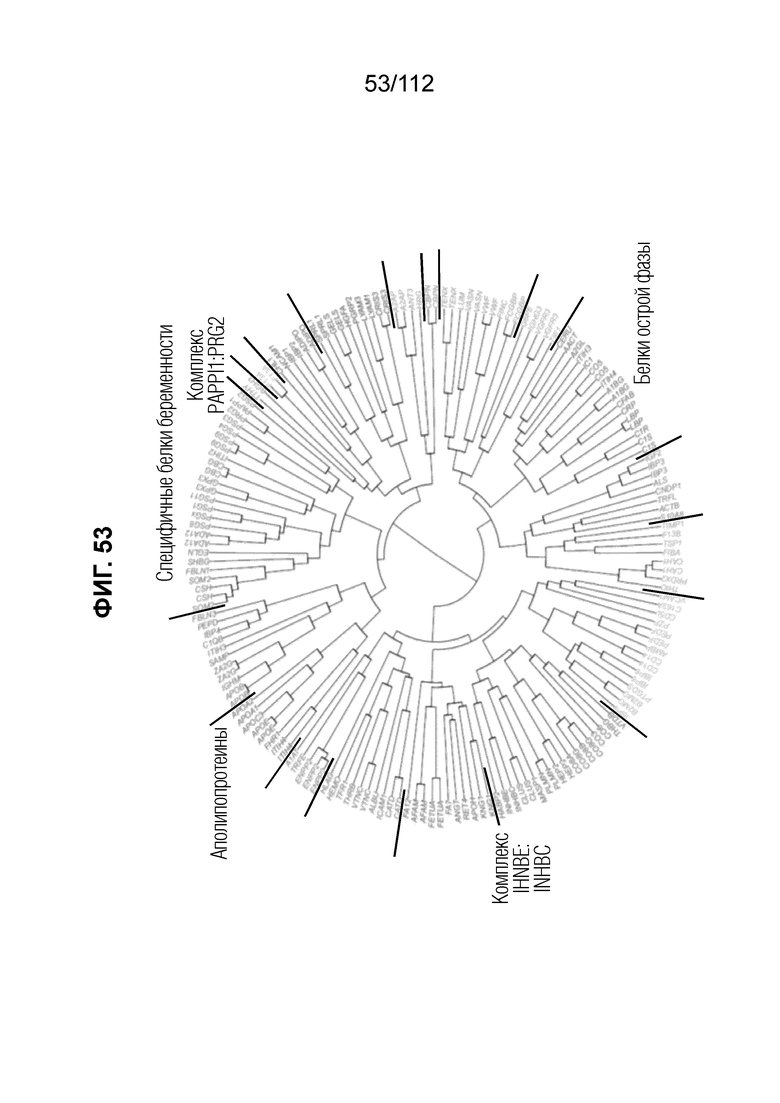
[0097] На фиг. 53 представлена иерархическая кластеризация коэффициентов ответа анализируемых веществ.
[0098] На фиг. 54 представлены дифференциально экспрессированные белки, которые функционируют во взаимодействиях внеклеточного матрикса.
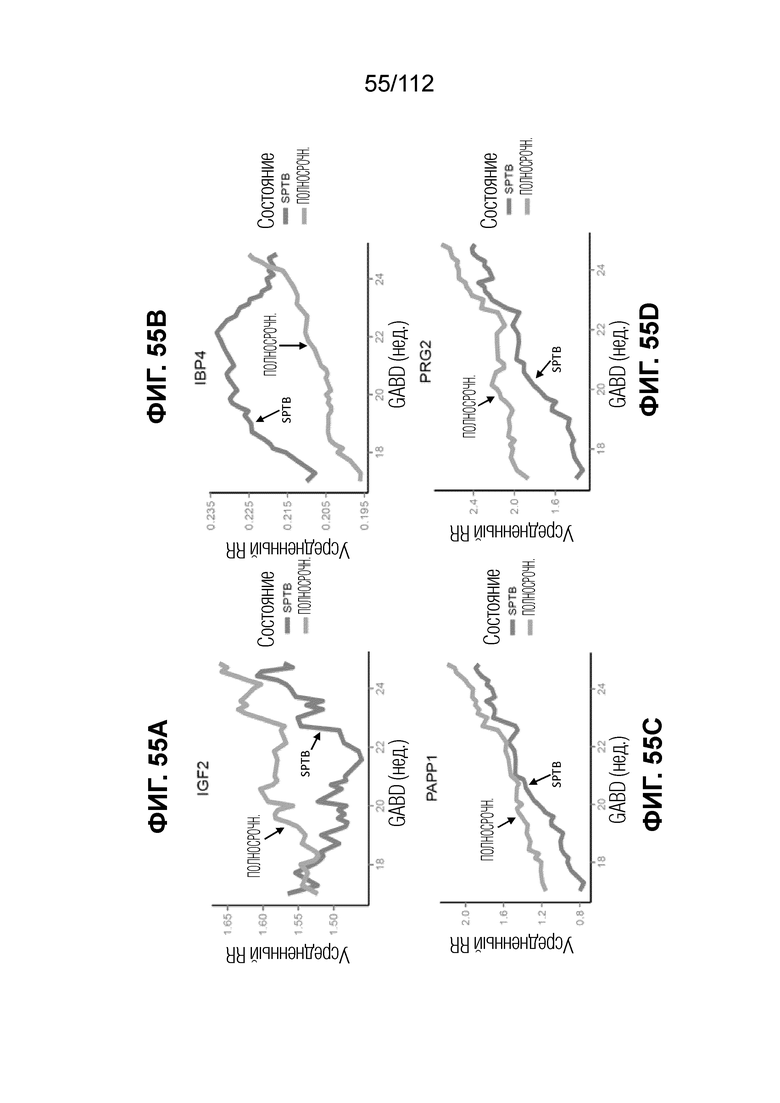
[0099] На фиг. 55 представлены кинетические графики дифференциально экспрессированных белков с функциями в пути IGF-2, которые демонстрируют максимальное разделение на 18 неделе.
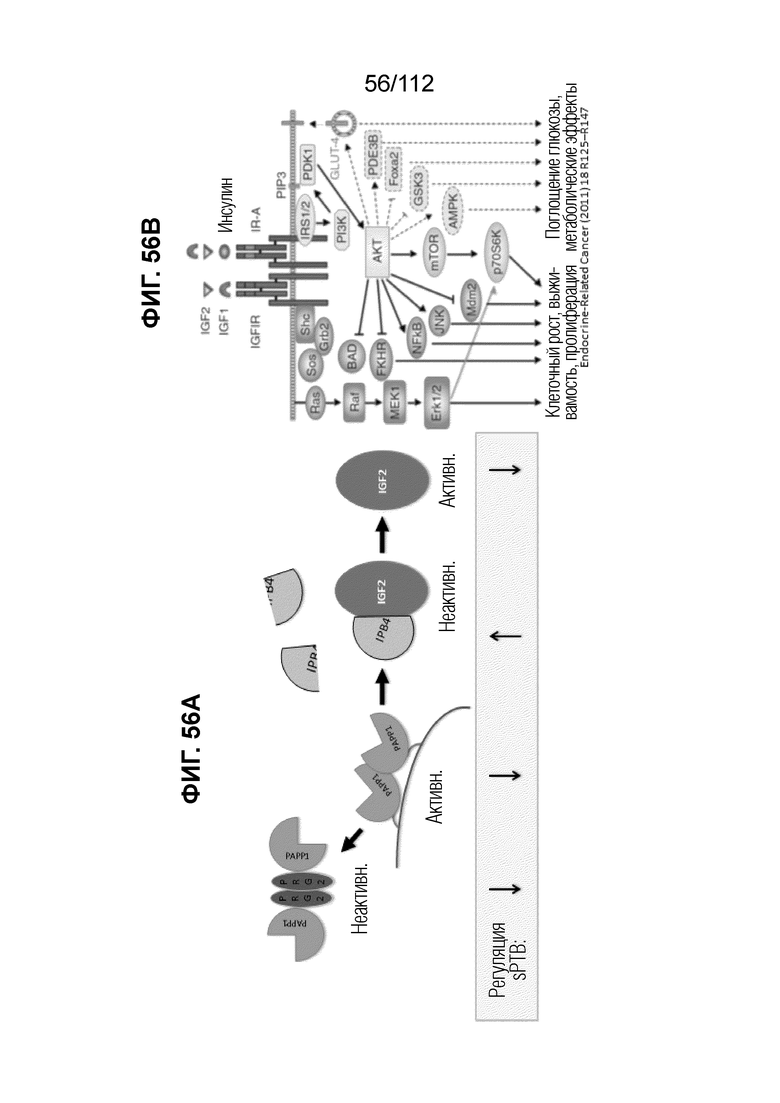
[00100] На фиг. 56A представлена схема взаимодействий между белками IGF-2, IBP4, PAPP1 и PRG2, влияющих на биодоступность этих белков при sPTB; на 56B представлена схема внутриклеточных сигналов, предпочтительно активируемых посредством связывания инсулина с IR-B и посредством связывания инсулина и IGF с их IR-A или IGF1R.
[00101] На фиг. 57 представлены кинетические графики дифференциально экспрессированных белков с функциями в равновесии метаболических гормонов.
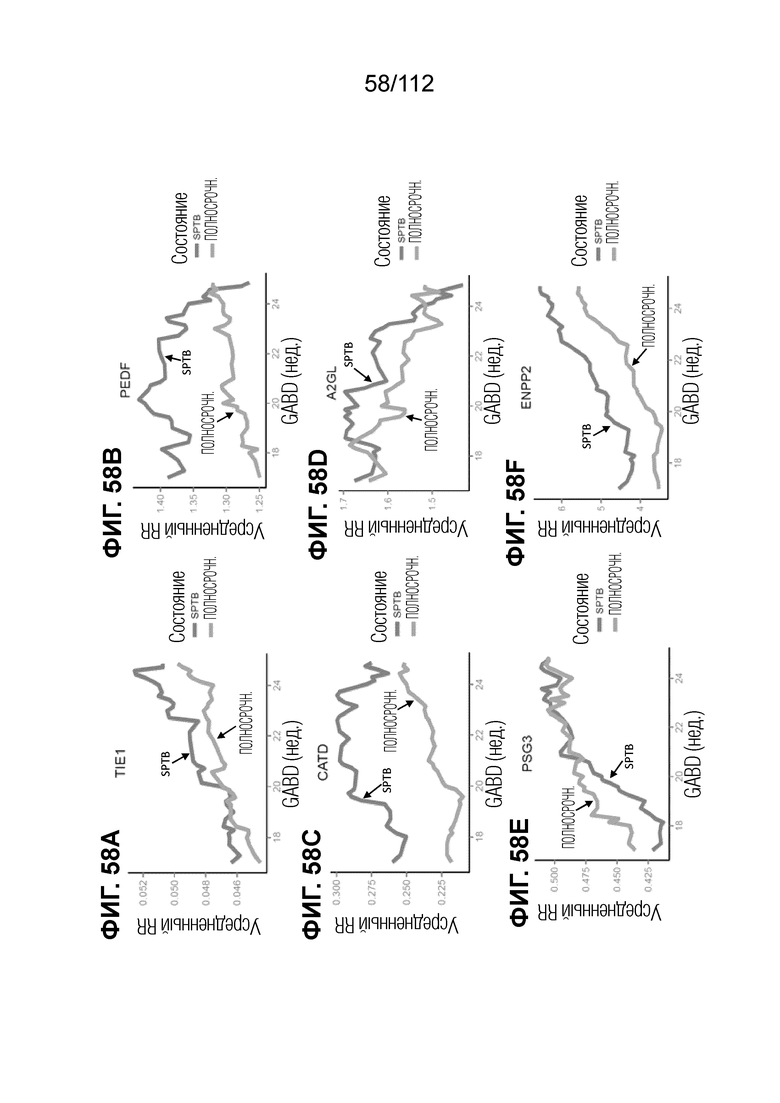
[00102] На фиг. 58 представлены кинетические графики дифференциально экспрессированных белков с функциями в ангиогенезе.
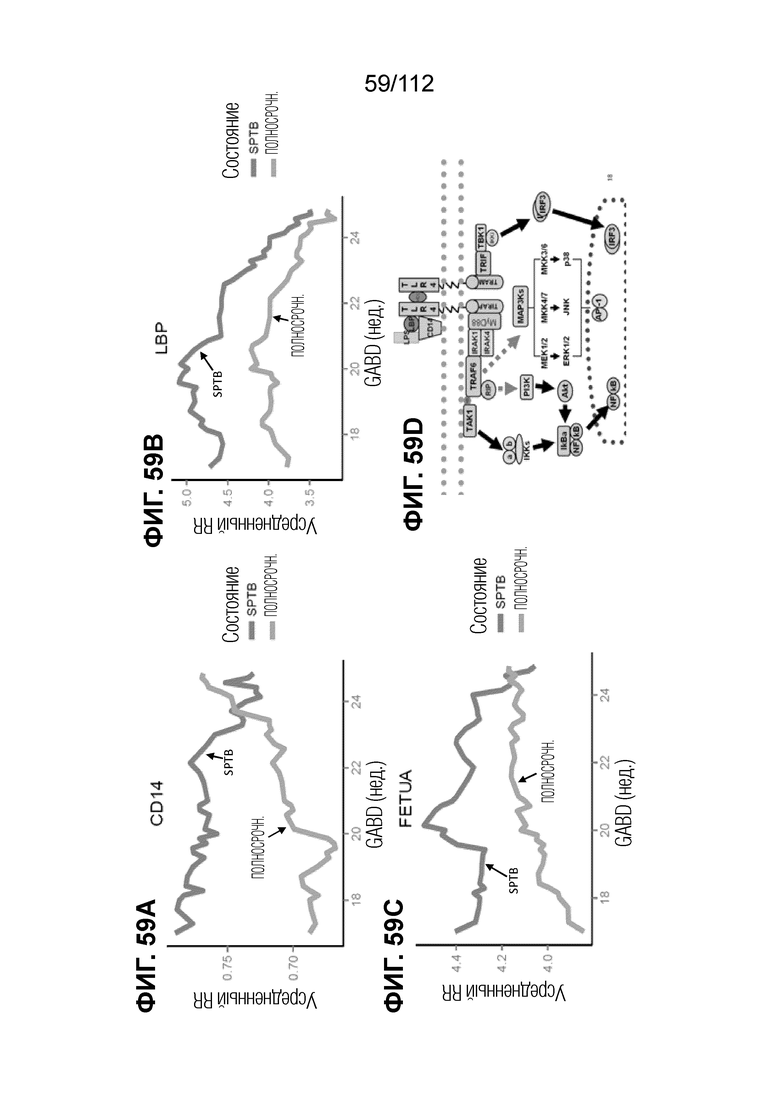
[00103] На фиг. 59 представлены кинетические графики дифференциально экспрессированных белков с функциями во врожденном иммунитете.
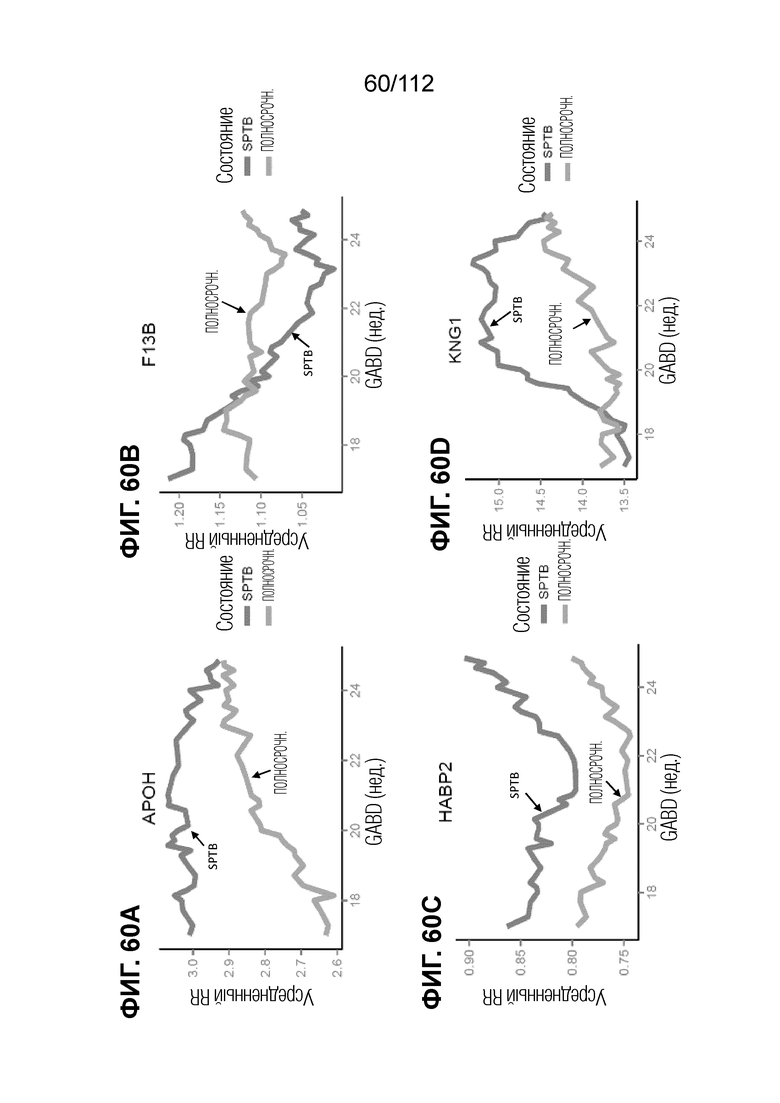
[00104] На фиг. 60 представлены кинетические графики дифференциально экспрессированных белков с функциями в коагуляции.
[00105] На фиг. 61 представлены кинетические графики дифференциально экспрессированных сывороточных/секретируемых белков.
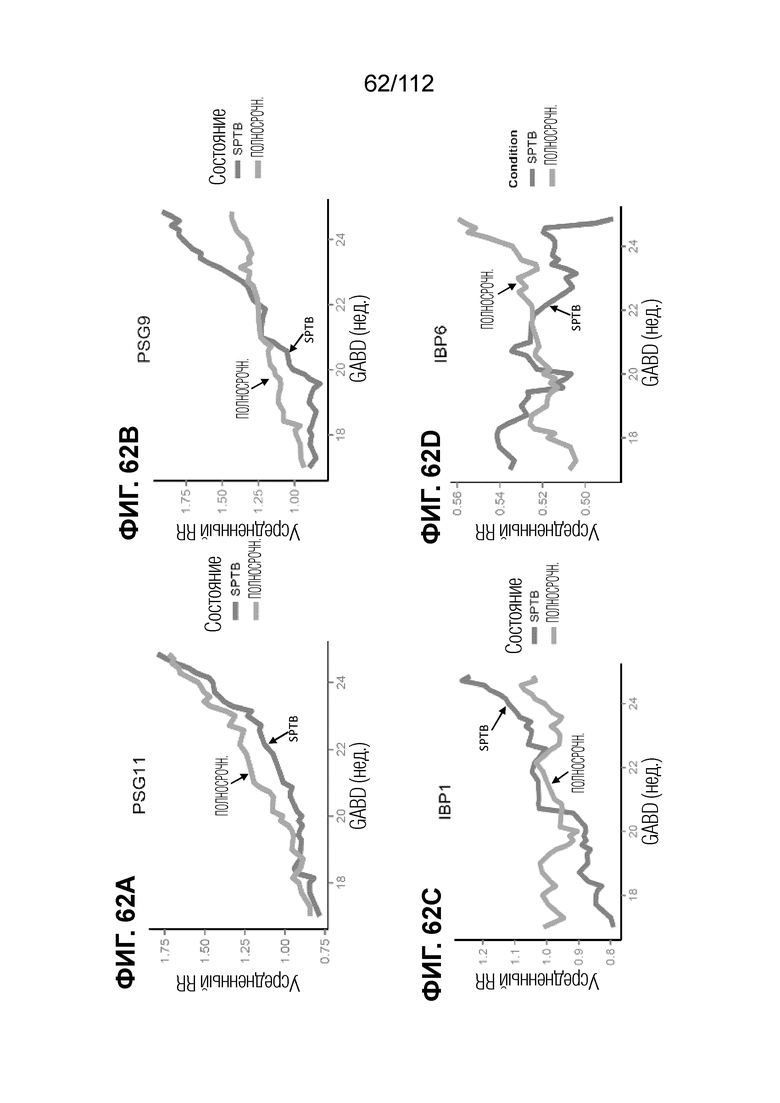
[00106] На фиг. 62 представлены кинетические графики дифференциально экспрессированных PSG/IBP.
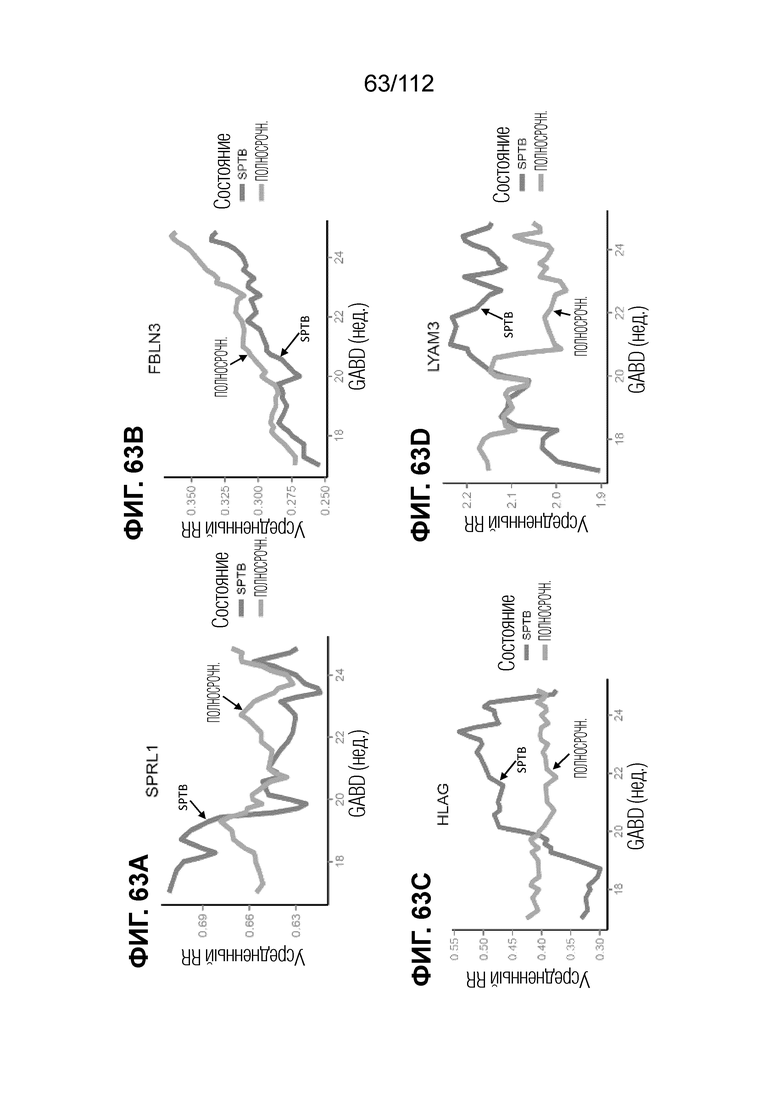
[00107] На фиг. 63 представлены кинетические графики дифференциально экспрессированных ECM/белков клеточной поверхности.
[00108] На фиг. 64 представлены кинетические графики дифференциально экспрессированных белков комплемента/белков острой фазы-1.
[00109] На фиг. 65 представлены кинетические графики дифференциально экспрессированных белков комплемента/белков острой фазы-2.
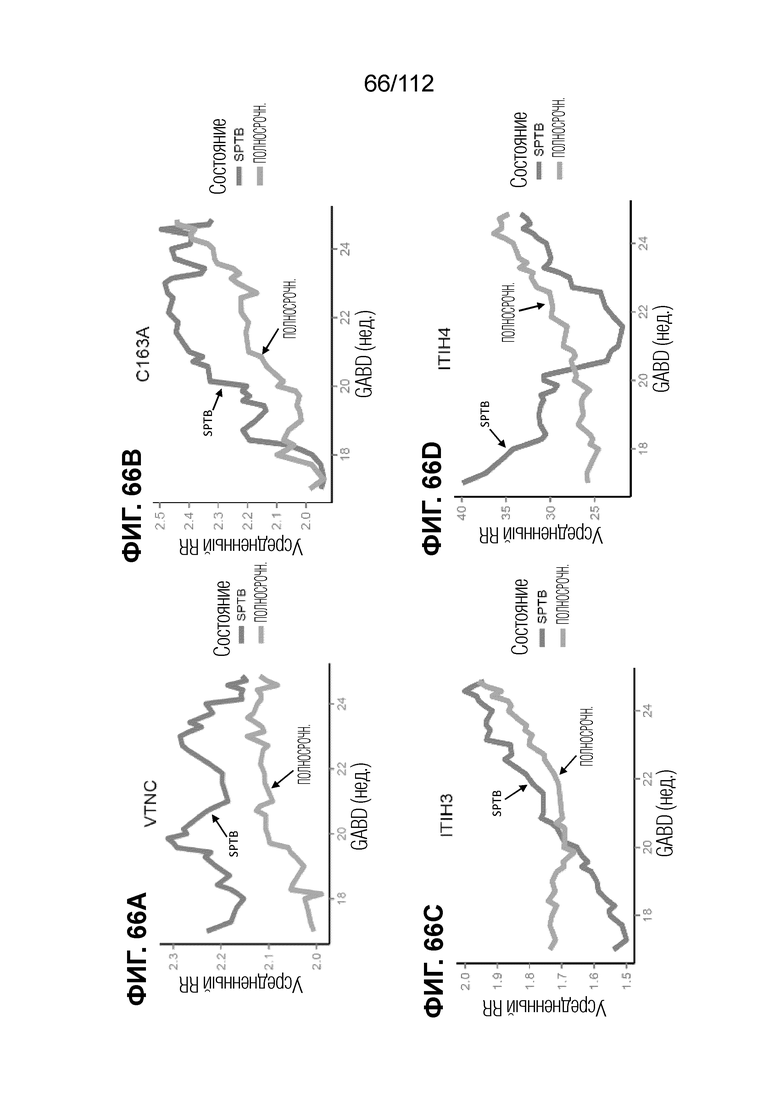
[00110] На фиг. 66 представлены кинетические графики дифференциально экспрессированных белков комплемента/белков острой фазы-3.
[00111] На фиг. 67 представлены кинетические графики дифференциально экспрессированных белков комплемента/белков острой фазы-4.
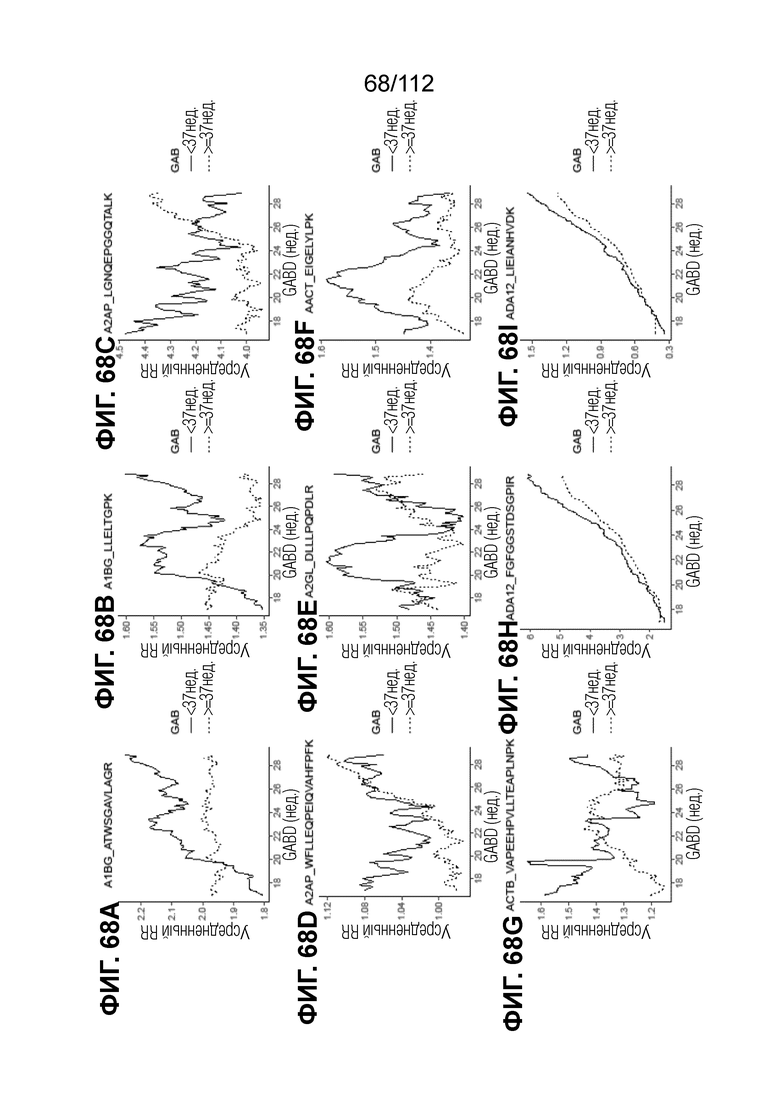
[00112] На фиг. 68 представлены кинетические графики для анализируемых веществ, которые определены в блоках с A до I, с данными о гестационном возрасте при взятии крови (GABD) от 17 недел 0 суток до 28 недель 6 суток.
[00113] На фиг. 69 представлены кинетические графики для анализируемых веществ, которые определены в блоках с A до I, с данными о GABD от 17 недель 0 суток до 28 недель 6 суток.
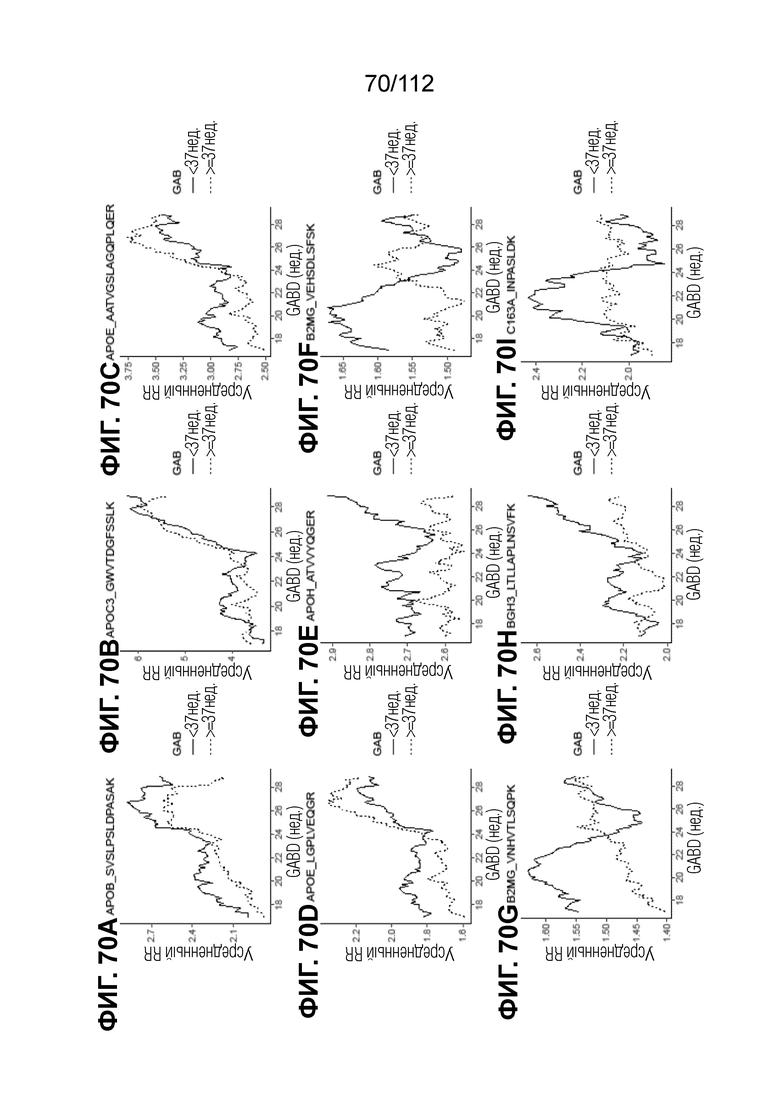
[00114] На фиг. 70 представлены кинетические графики для анализируемых веществ, которые определены в блоках с A до I, с данными о GABD от 17 недель 0 суток до 28 недель 6 суток.
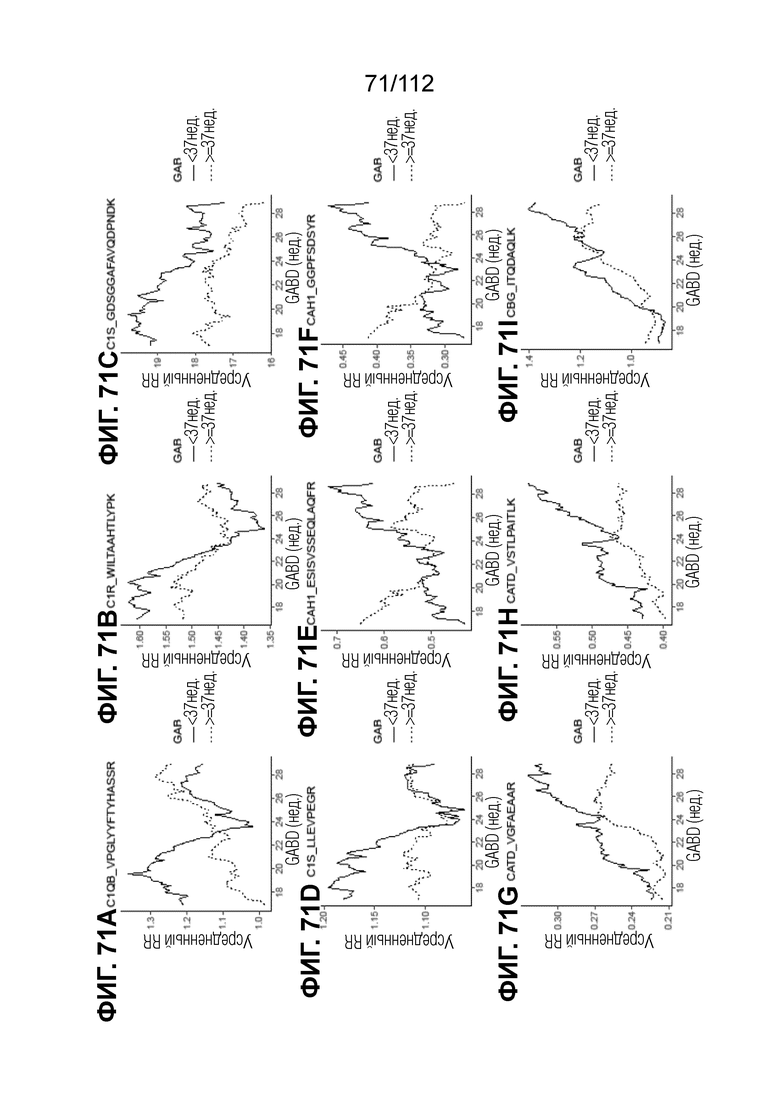
[00115] На фиг. 71 представлены кинетические графики для анализируемых веществ, которые определены в блоках с A до I, с данными о GABD от 17 недель 0 суток до 28 недель 6 суток.
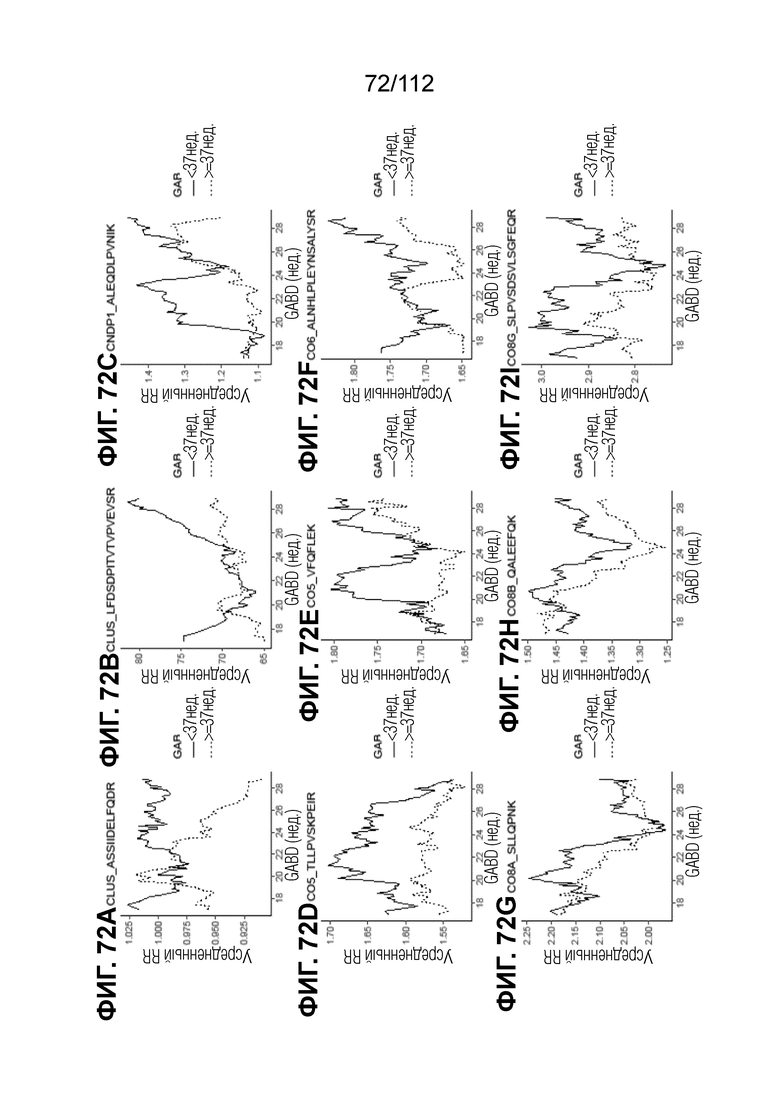
[00116] На фиг. 72 представлены кинетические графики для анализируемых веществ, которые определены в блоках с A до I, с данными о GABD от 17 недель 0 суток до 28 недель 6 суток.
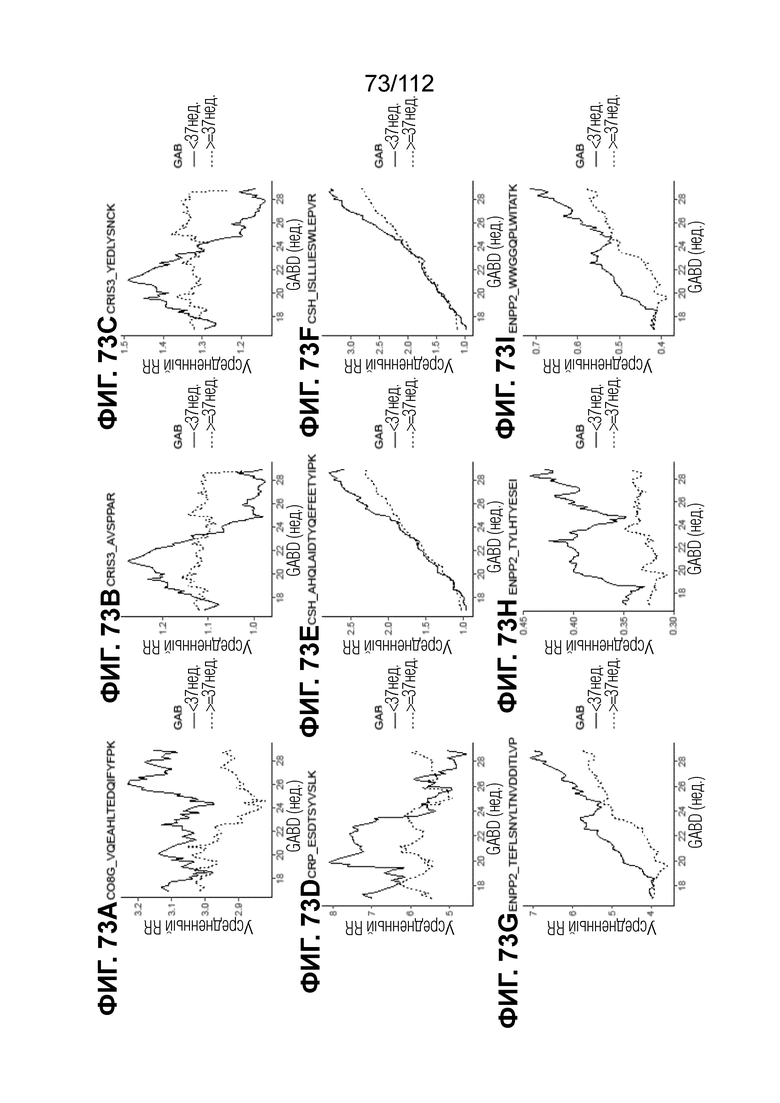
[00117] На фиг. 73 представлены кинетические графики для анализируемых веществ, которые определены в блоках с A до I, с данными о GABD от 17 недель 0 суток до 28 недель 6 суток.
[00118] На фиг. 74 представлены кинетические графики для анализируемых веществ, которые определены в блоках с A до I, с данными о GABD от 17 недель 0 суток до 28 недель 6 суток.
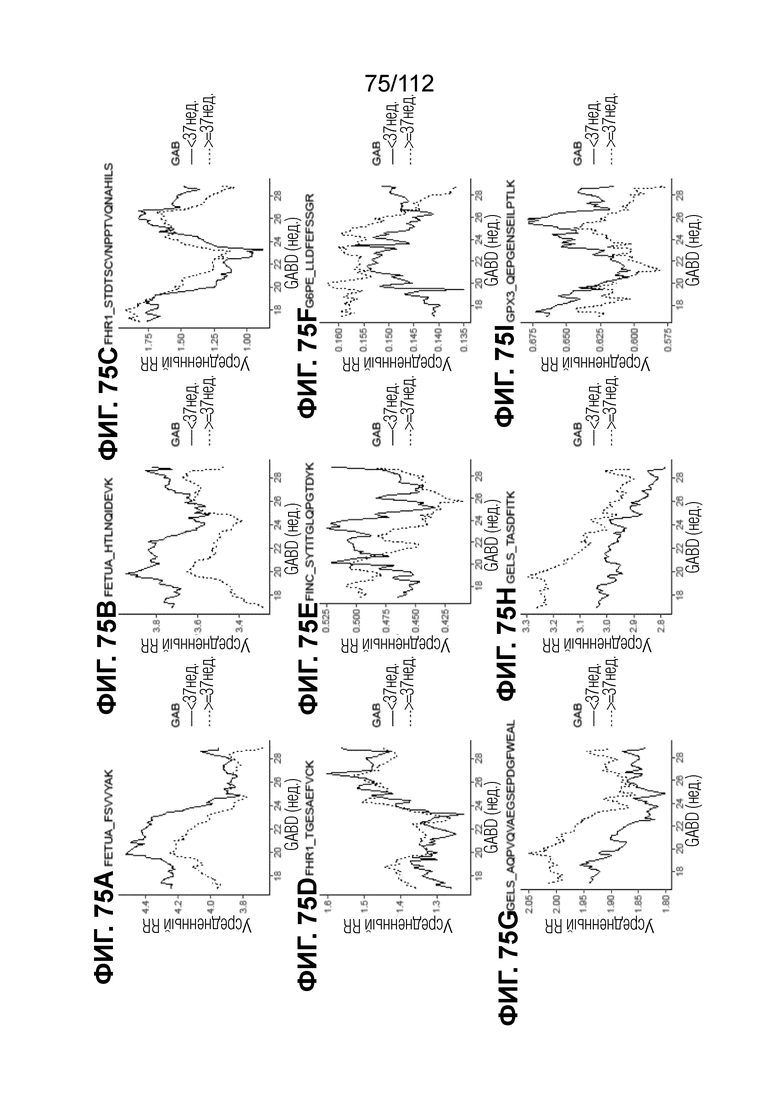
[00119] На фиг. 75 представлены кинетические графики для анализируемых веществ, которые определены в блоках с A до I, с данными о GABD от 17 недель 0 суток до 28 недель 6 суток.
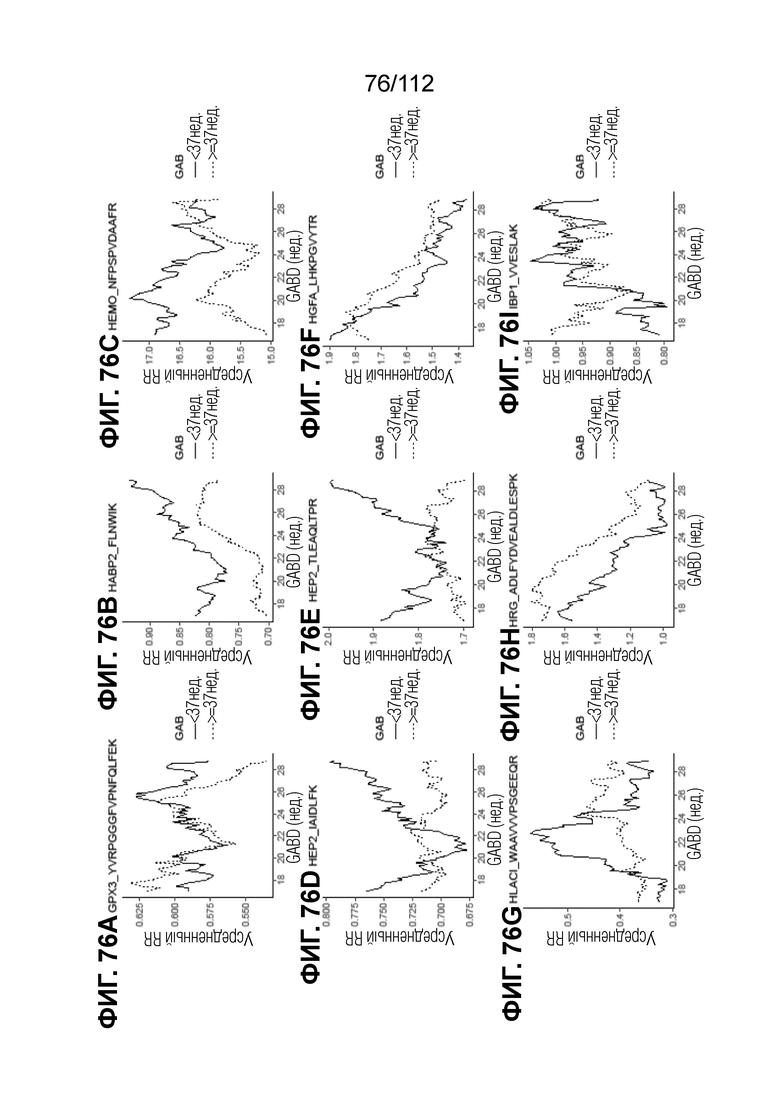
[00120] На фиг. 76 представлены кинетические графики для анализируемых веществ, которые определены в блоках с A до I, с данными о GABD от 17 недель 0 суток до 28 недель 6 суток.
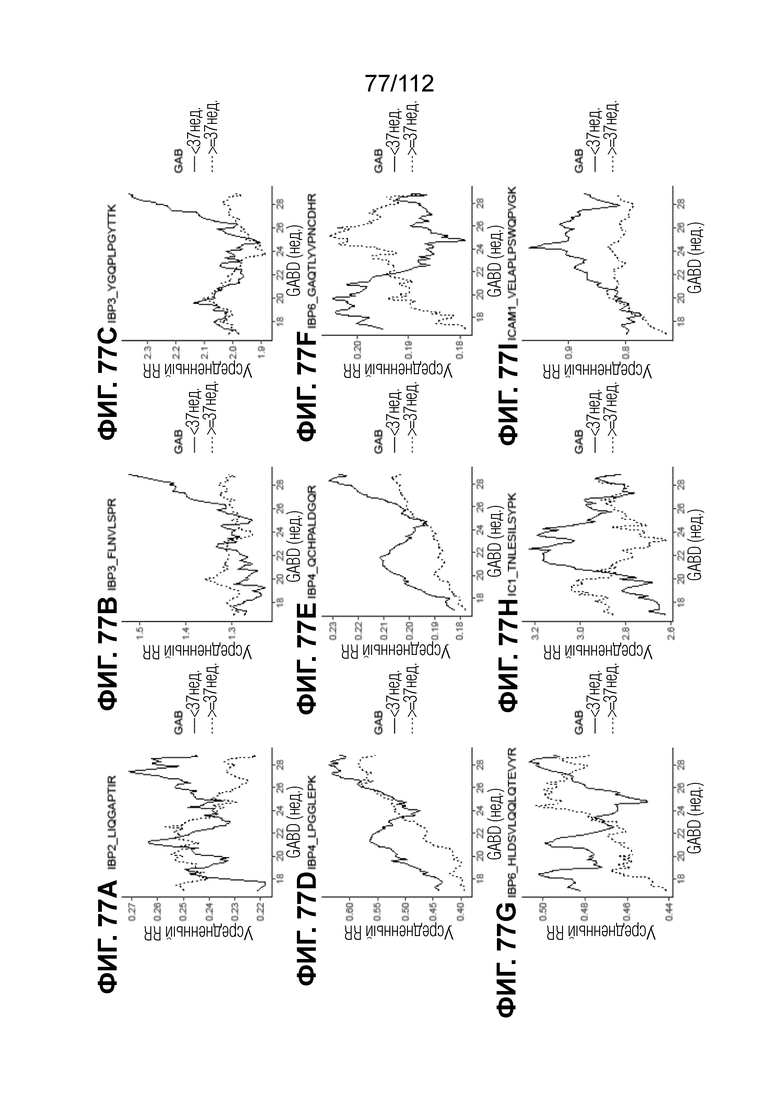
[00121] На фиг. 77 представлены кинетические графики для анализируемых веществ, которые определены в блоках с A до I, с данными о GABD от 17 недель 0 суток до 28 недель 6 суток.
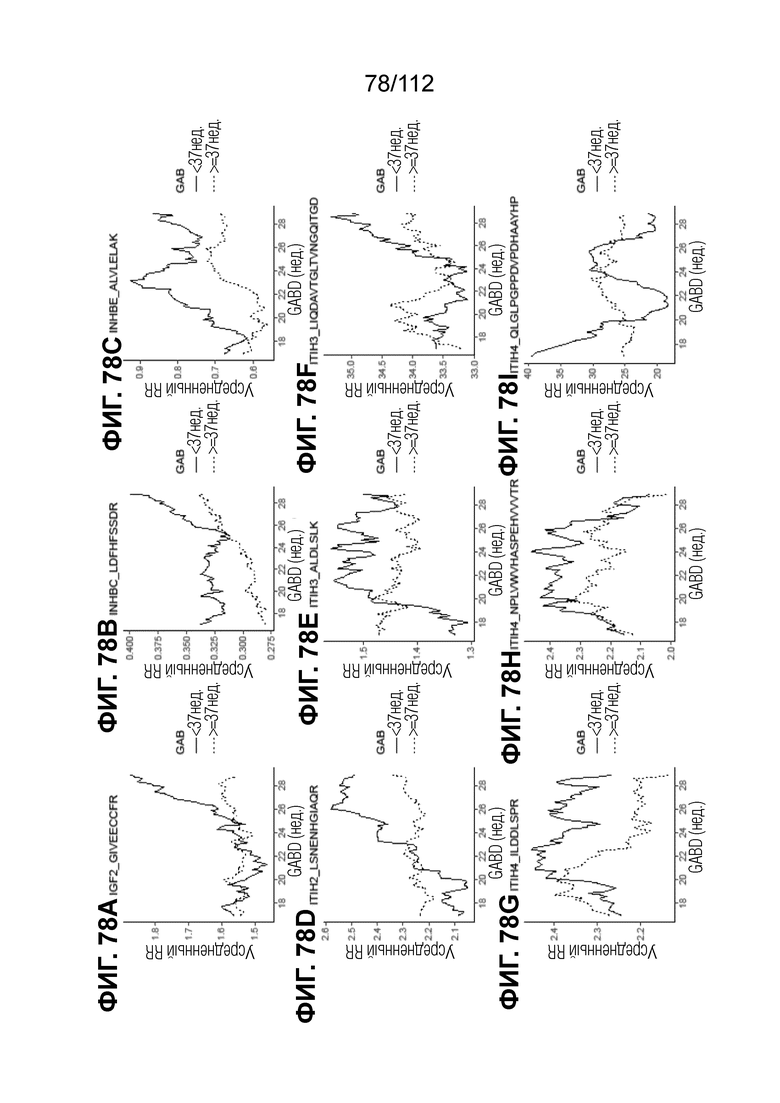
[00122] На фиг. 78 представлены кинетические графики для анализируемых веществ, которые определены в блоках с A до I, с данными о GABD от 17 недель 0 суток до 28 недель 6 суток.
[00123] На фиг. 79 представлены кинетические графики для анализируемых веществ, которые определены в блоках с A до I, с данными о GABD от 17 недель 0 суток до 28 недель 6 суток.
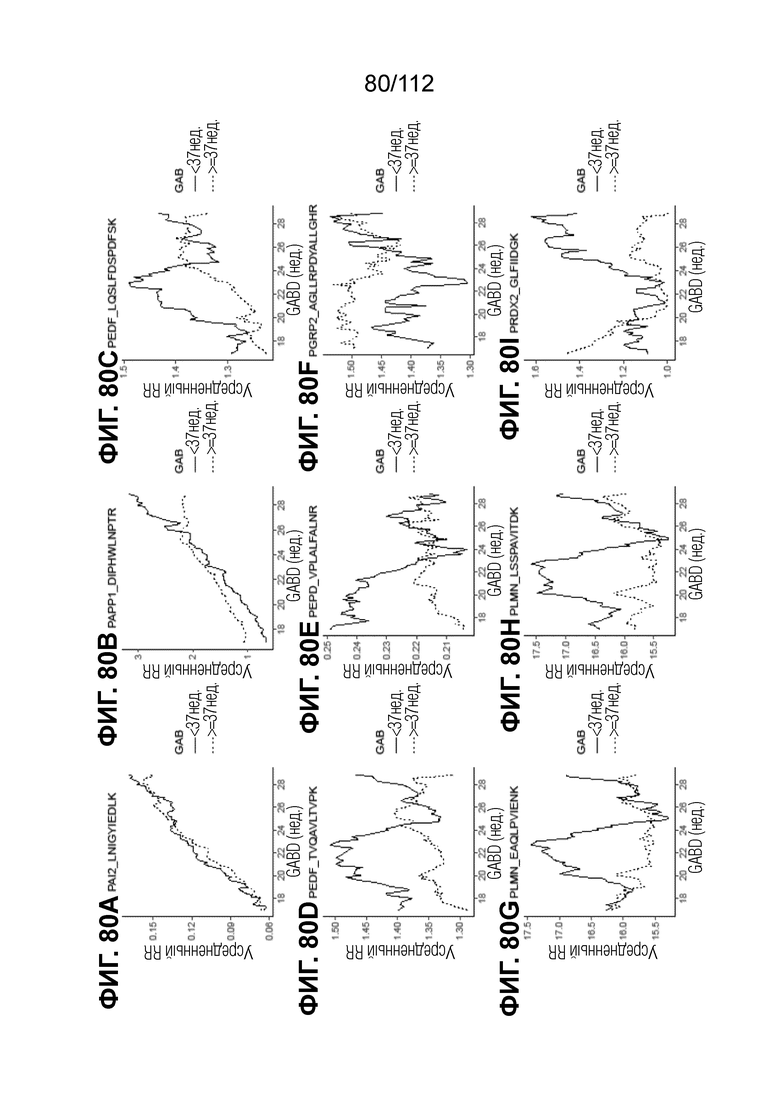
[00124] На фиг. 80 представлены кинетические графики для анализируемых веществ, которые определены в блоках с A до I, с данными о GABD от 17 недель 0 суток до 28 недель 6 суток.
[00125] На фиг. 81 представлены кинетические графики для анализируемых веществ, которые определены в блоках с A до I, с данными о GABD от 17 недель 0 суток до 28 недель 6 суток.
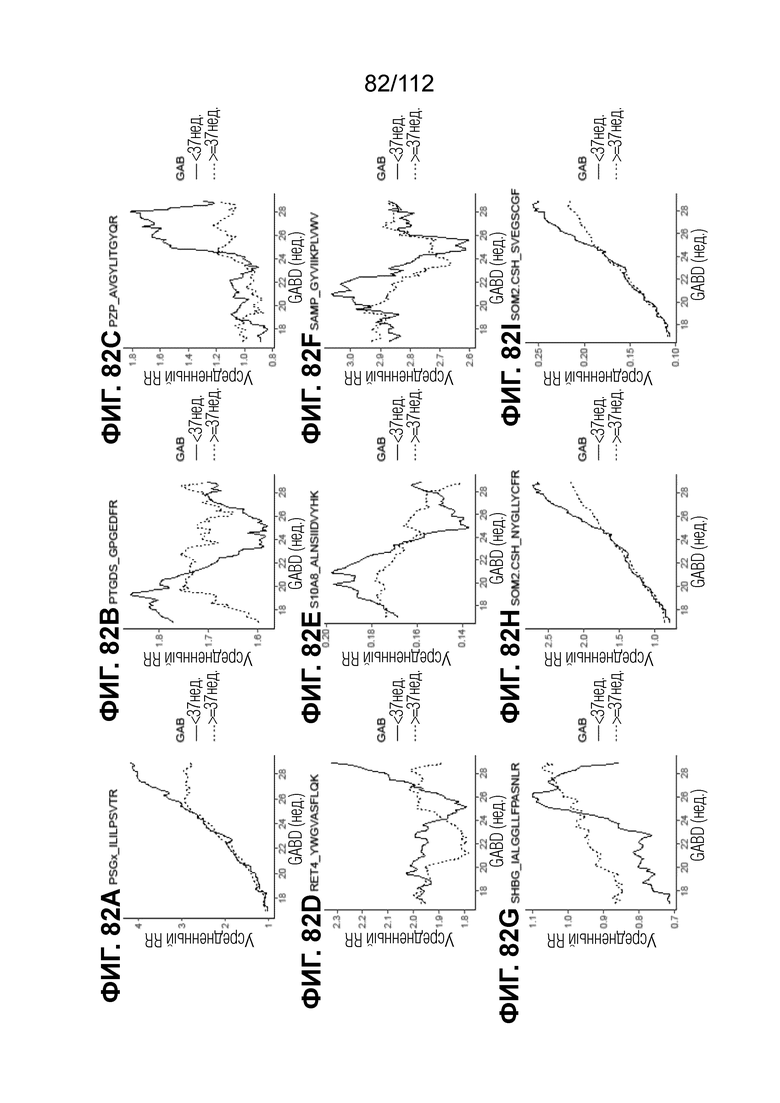
[00126] На фиг. 82 представлены кинетические графики для анализируемых веществ, которые определены в блоках с A до I, с данными от 17 недель 0 суток до 28 недель 6 суток.
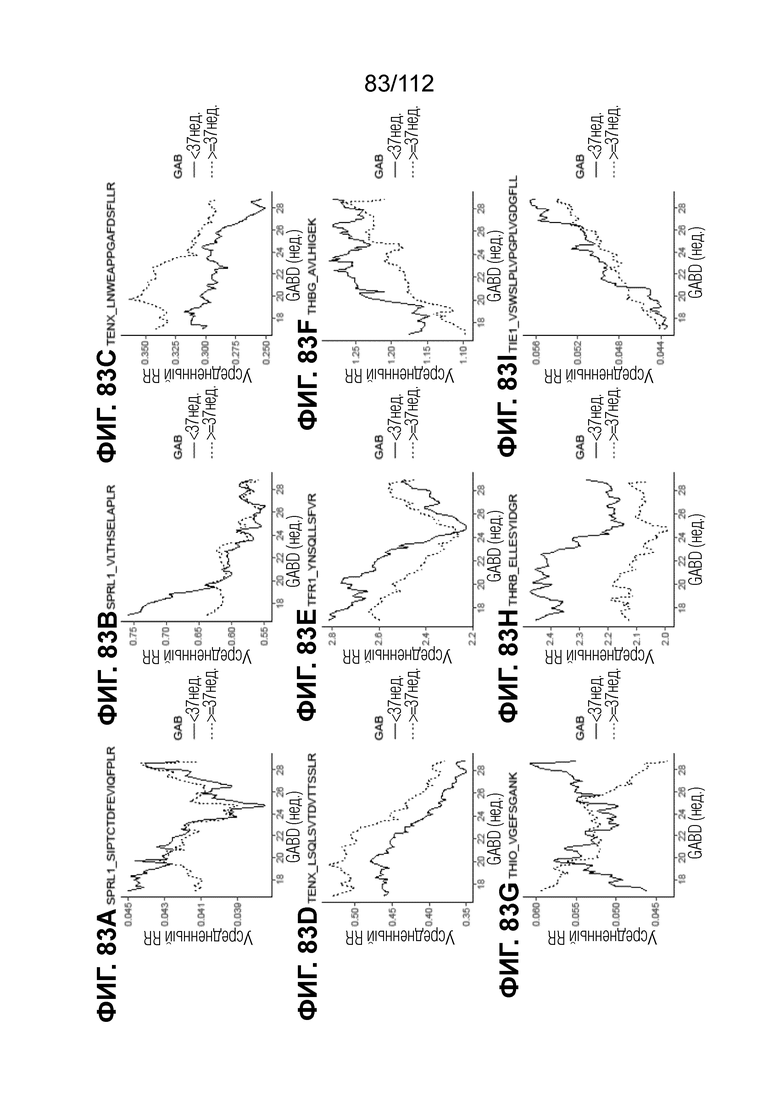
[00127] На фиг. 83 представлены кинетические графики для анализируемых веществ, которые определены в блоках с A до I, с данными о GABD от 17 недель 0 суток до 28 недель 6 суток.
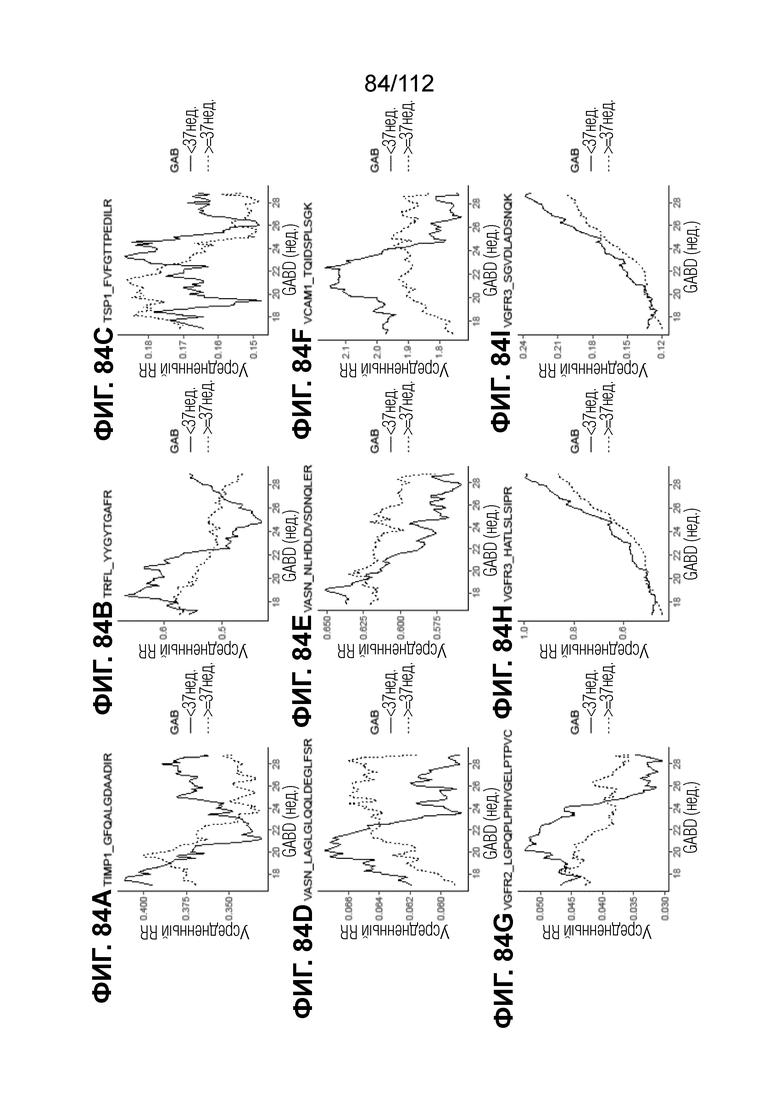
[00128] На фиг. 84 представлены кинетические графики для анализируемых веществ, которые определены в блоках с A до I, с данными о GABD от 17 недель 0 суток до 28 недель 6 суток.
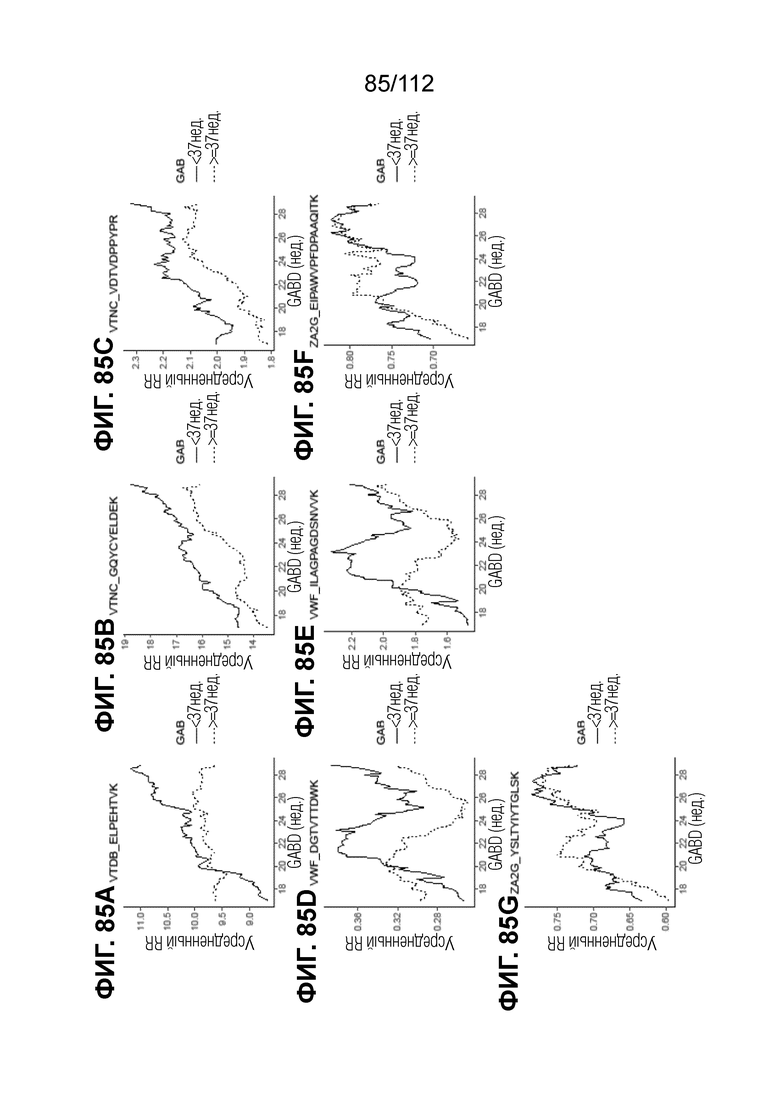
[00129] На фиг. 85 представлены кинетические графики для переходов пептидов, которые определены в блоках с A до G, с данными о GABD от 17 недель 0 суток до 28 недель 6 суток.
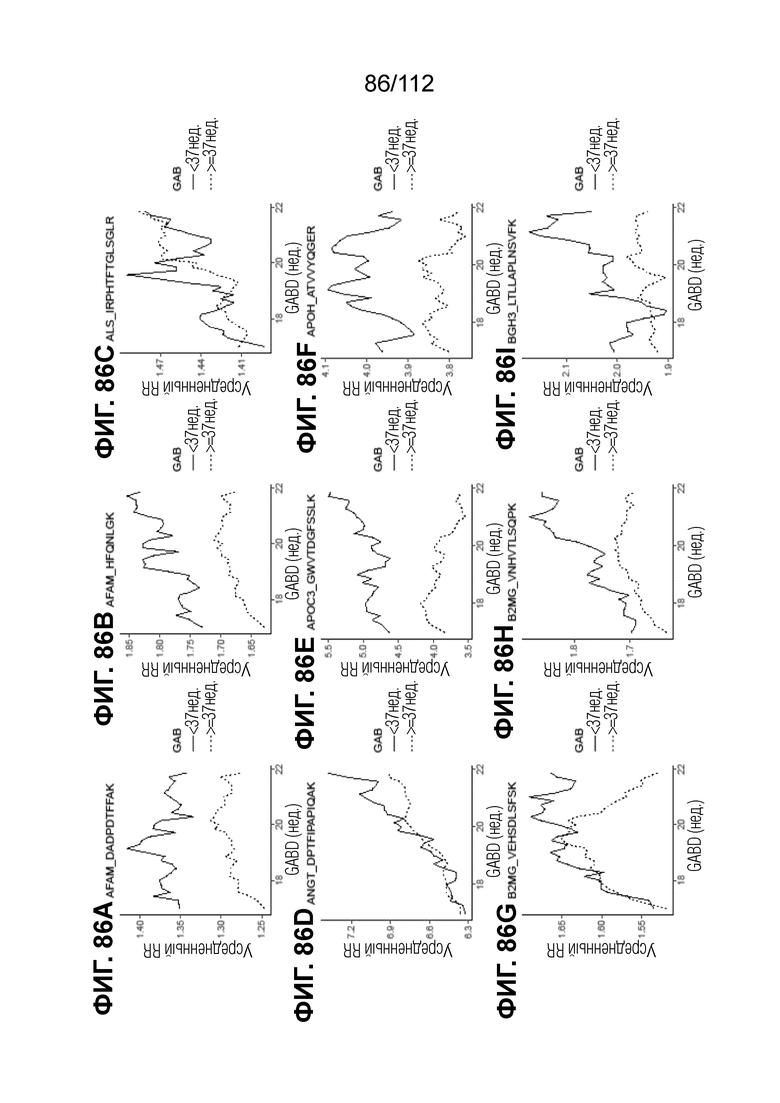
[00130] На фиг. 86 представлены кинетические графики для переходов пептидов, которые определены в блоках с A до I, с использованием порога гестационного возраста при рождении <37 0/7 в сравнении с≥37 0/7 недель.
[00131] На фиг. 87 представлены кинетические графики для переходов пептидов, которые определены в блоках с A до I, с использованием порога гестационного возраста при рождении <37 0/7 в сравнении с≥37 0/7 недель.
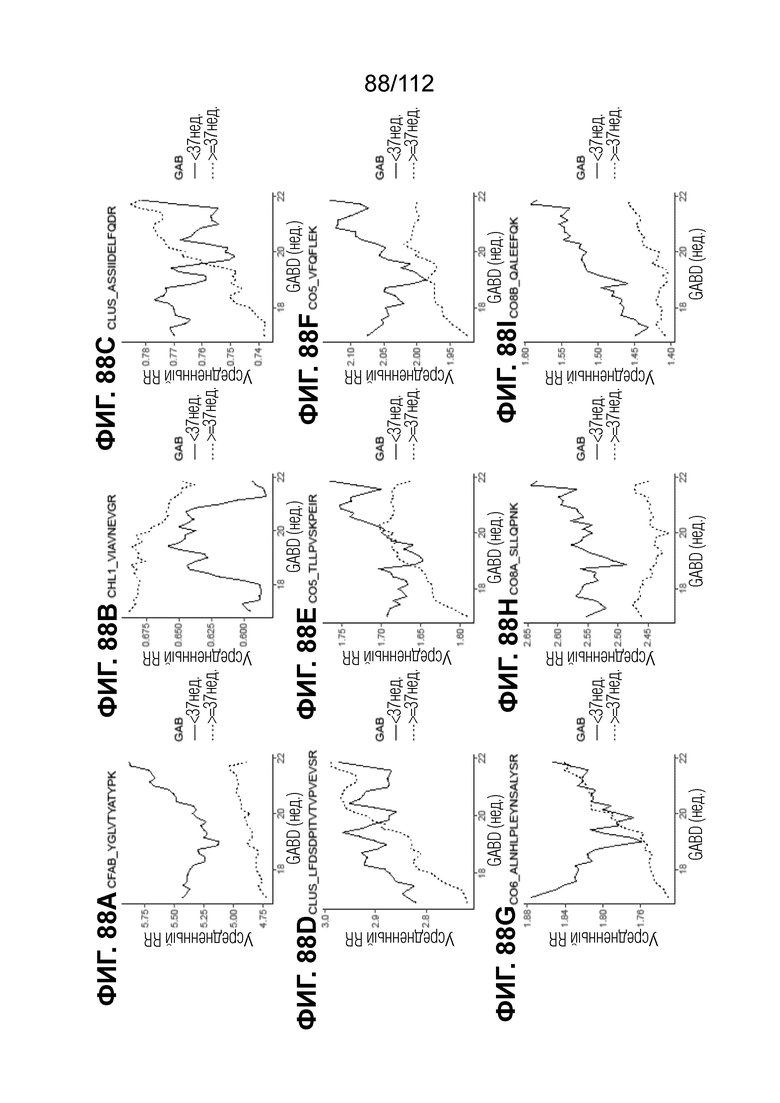
[00132] На фиг. 88 представлены кинетические графики для переходов пептидов, которые определены в блоках с A до I, с использованием порога гестационного возраста при рождении <37 0/7 в сравнении с≥37 0/7 недель.
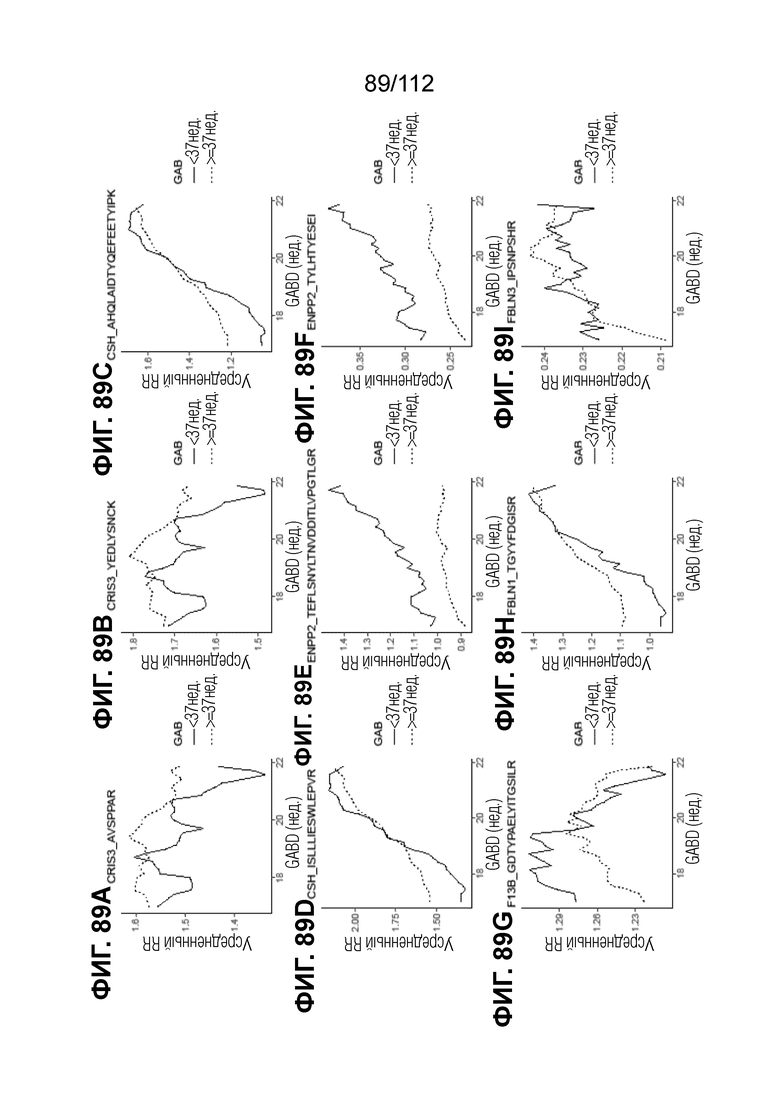
[00133] На фиг. 89 представлены кинетические графики для переходов пептидов, которые определены в блоках с A до I, с использованием порога гестационного возраста при рождении <37 0/7 в сравнении с≥37 0/7 недель.
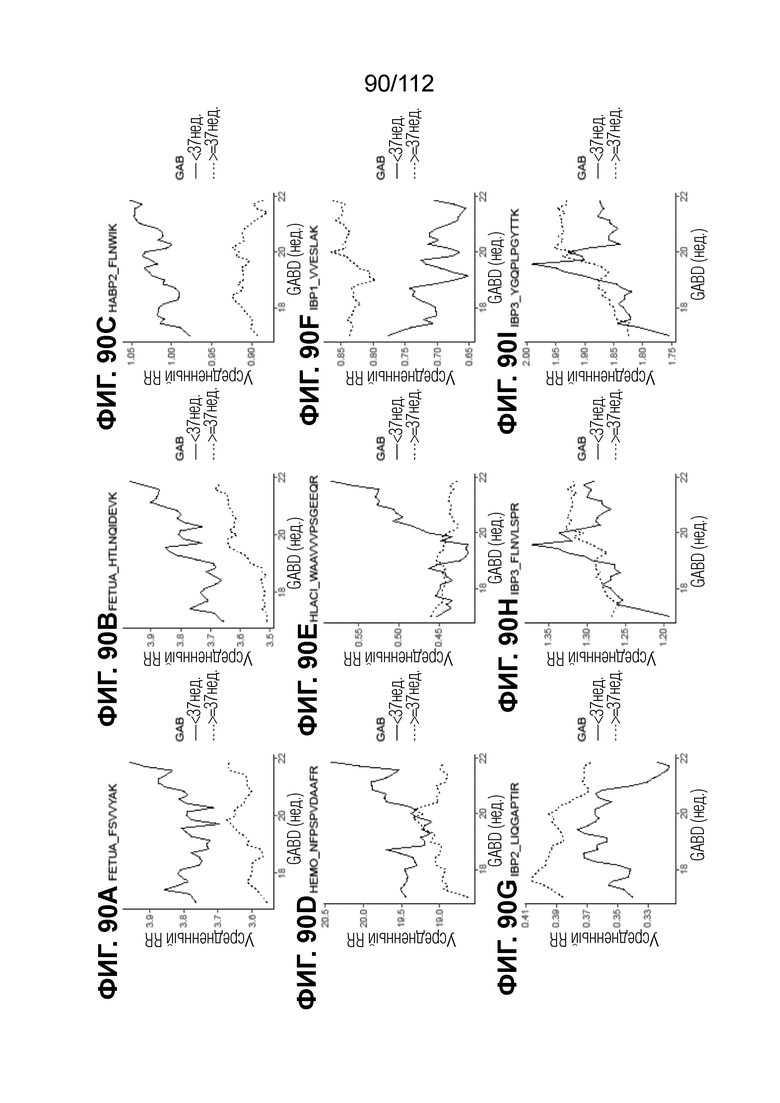
[00134] На фиг. 90 представлены кинетические графики для переходов пептидов, которые определены в блоках с A до I, с использованием порога гестационного возраста при рождении <37 0/7 в сравнении с≥37 0/7 недель.
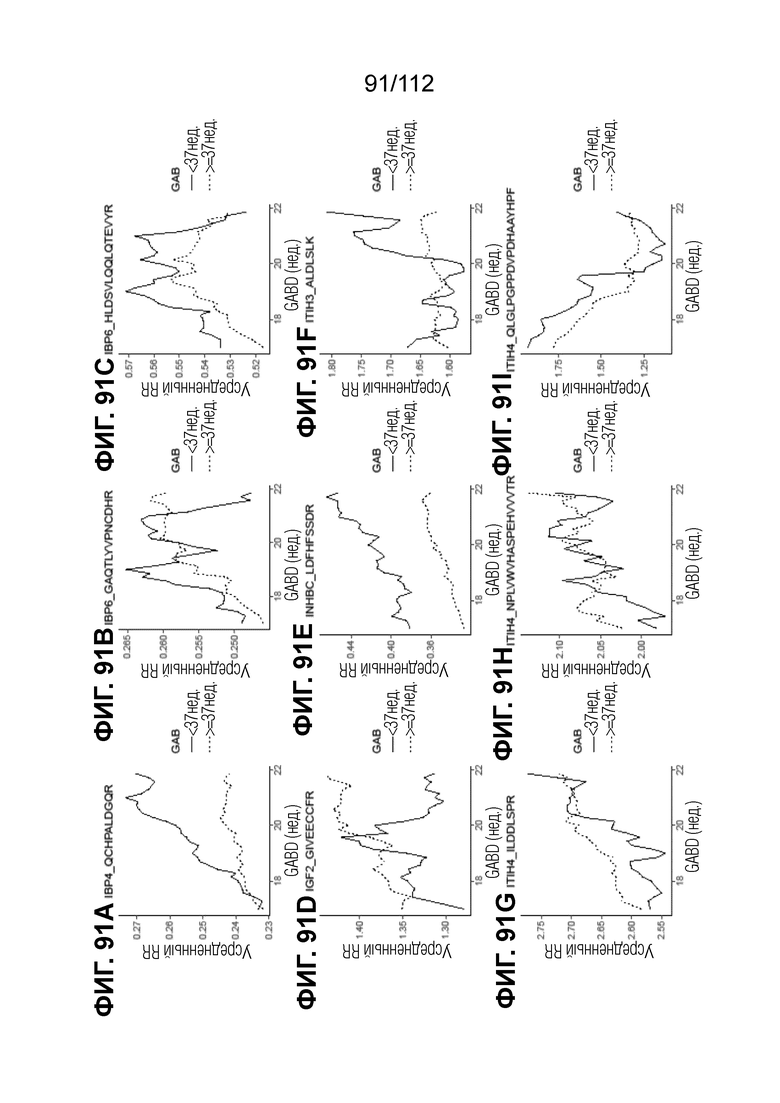
[00135] На фиг. 91 представлены кинетические графики для переходов пептидов, которые определены в блоках с A до I, с использованием порога гестационного возраста при рождении <37 0/7 в сравнении с≥37 0/7 недель.
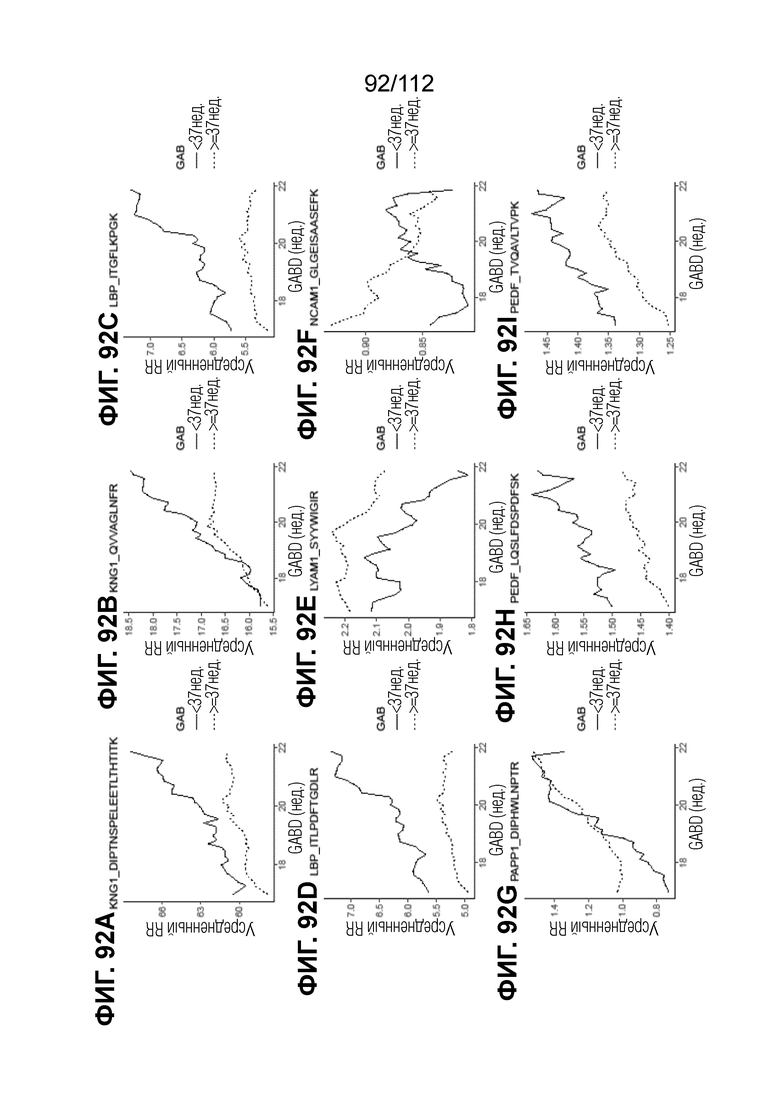
[00136] На фиг. 92 представлены кинетические графики для переходов пептидов, которые определены в блоках с A до I, с использованием порога гестационного возраста при рождении <37 0/7 в сравнении с≥37 0/7 недель.
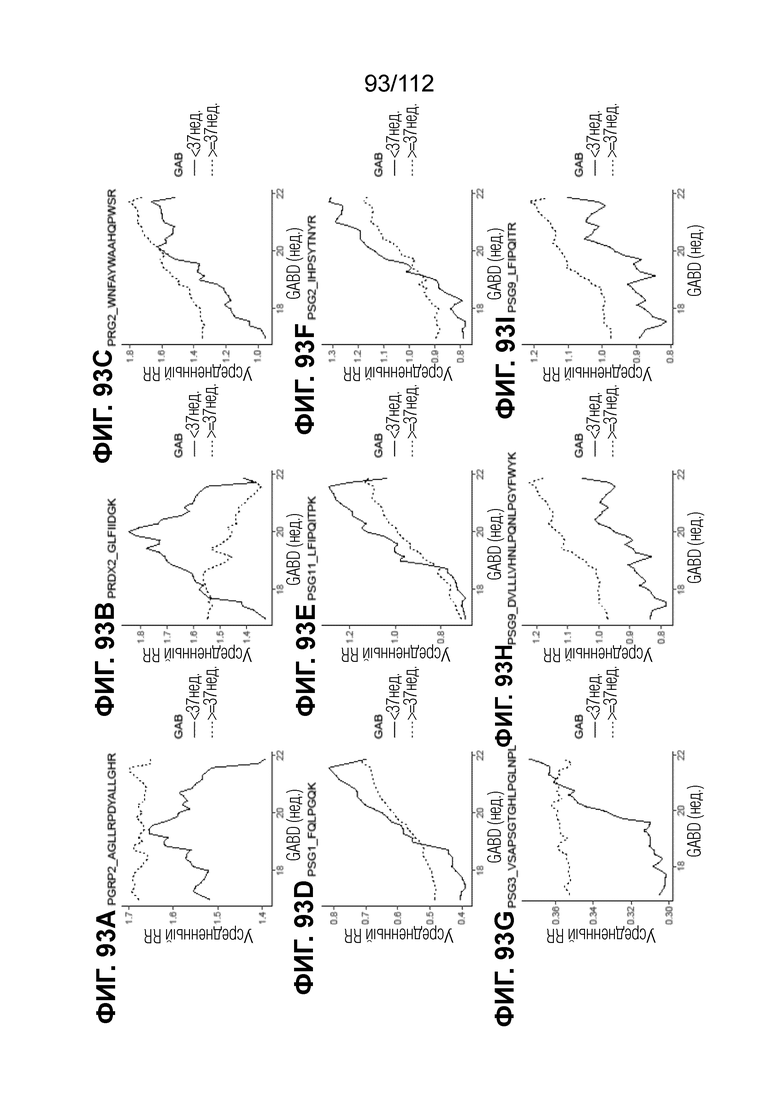
[00137] На фиг. 93 представлены кинетические графики для переходов пептидов, которые определены в блоках с A до I, с использованием порога гестационного возраста при рождении <37 0/7 в сравнении с≥37 0/7 недель.
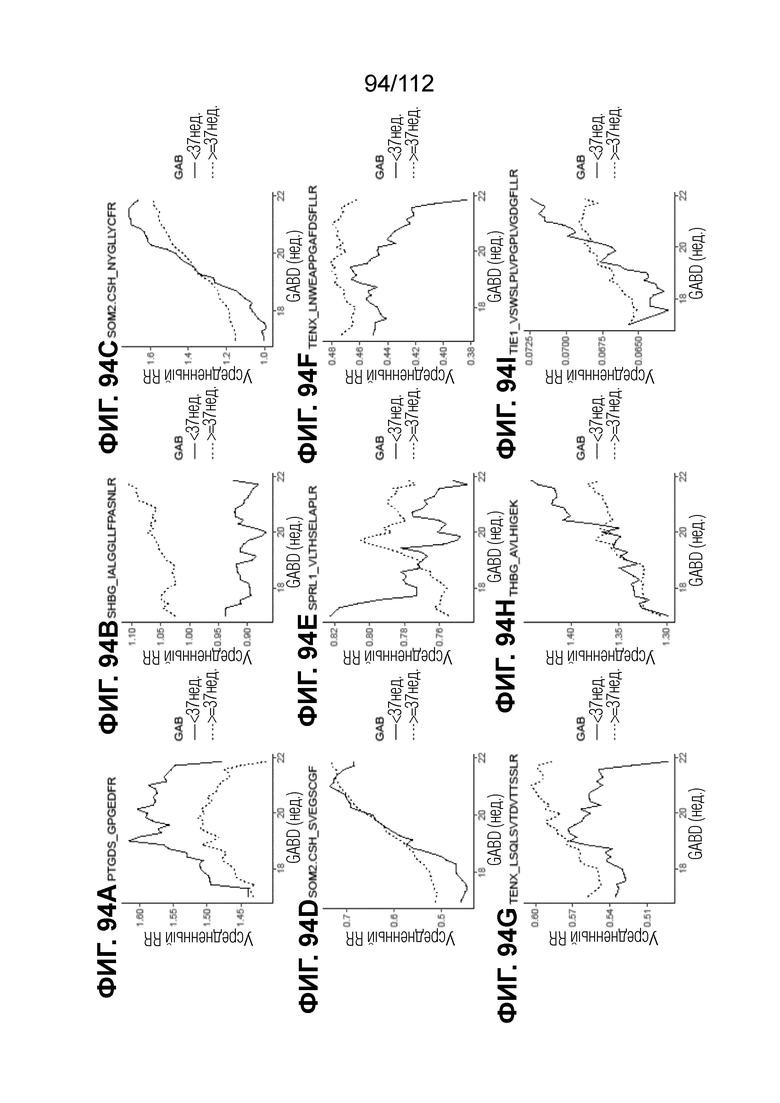
[00138] На фиг. 94 представлены кинетические графики для переходов пептидов, которые определены в блоках с A до I, с использованием порога гестационного возраста при рождении <37 0/7 в сравнении с≥37 0/7 недель.
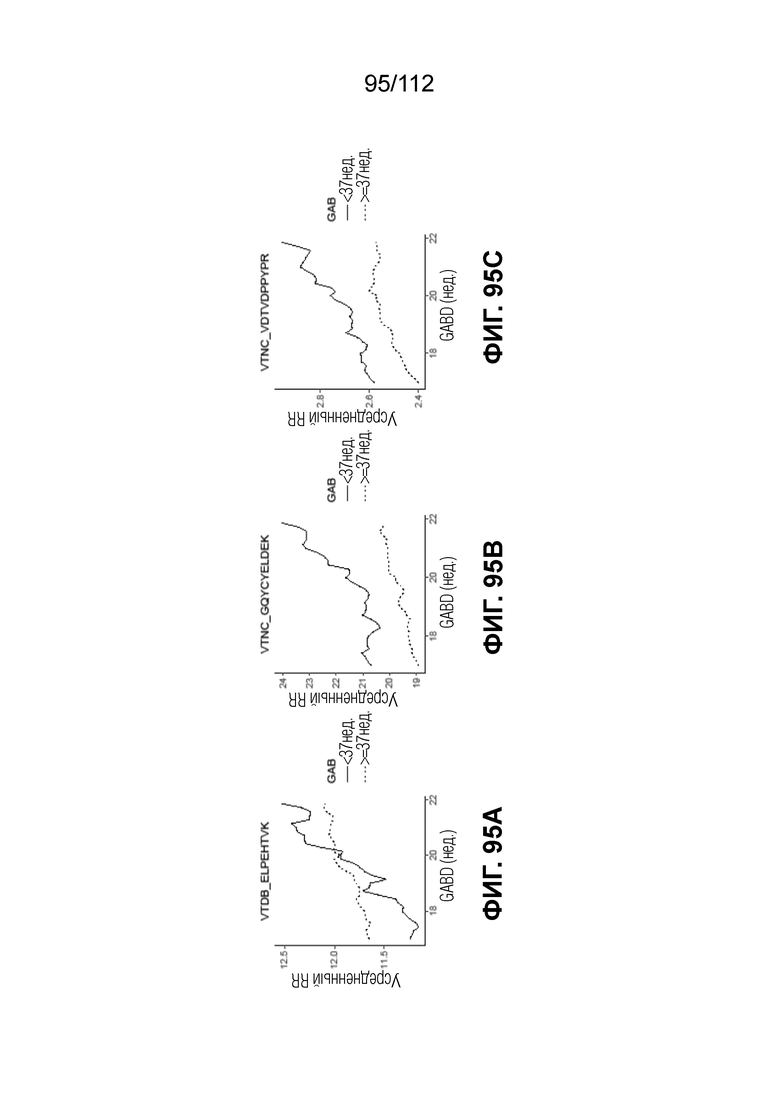
[00139] На фиг. 95 представлены кинетические графики для переходов пептидов, которые определены в блоках с A до C, с использованием порога гестационного возраста при рождении <37 0/7 в сравнении с≥37 0/7 недель.
[00140] На фиг. 96 представлены кинетические графики для переходов пептидов, которые определены в блоках с A до I, с использованием порога гестационного возраста при рождении <35 0/7 в сравнении с≥35 0/7 недель.
[00141] На фиг. 97 представлены кинетические графики для переходов пептидов, которые определены в блоках с A до I, с использованием порога гестационного возраста при рождении <35 0/7 в сравнении с≥35 0/7 недель.
[00142] На фиг. 98 представлены кинетические графики для переходов пептидов, которые определены в блоках с A до I, с использованием порога гестационного возраста при рождении <35 0/7 в сравнении с≥35 0/7 недель.
[00143] На фиг. 99 представлены кинетические графики для переходов пептидов, которые определены в блоках с A до I, с использованием порога гестационного возраста при рождении <35 0/7 в сравнении с≥35 0/7 недель.
[00144] На фиг. 100 представлены кинетические графики для переходов пептидов, которые определены в блоках с A до I, с использованием порога гестационного возраста при рождении <35 0/7 в сравнении с≥35 0/7 недель.
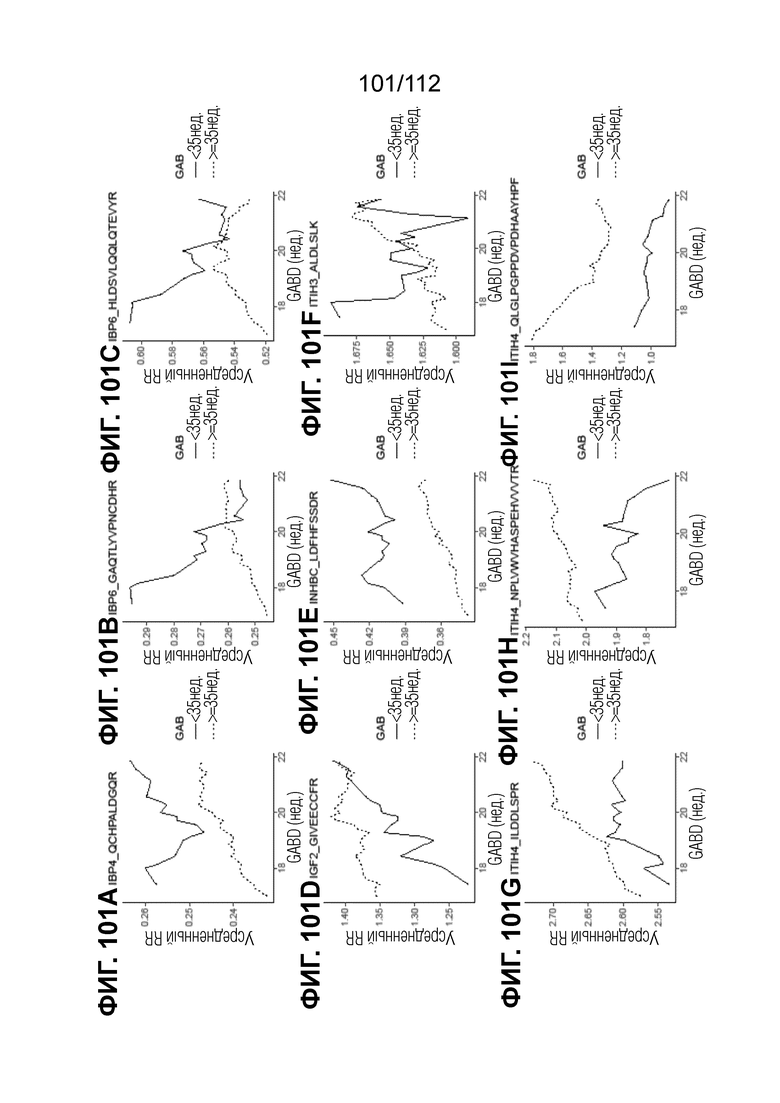
[00145] На фиг. 101 представлены кинетические графики для переходов пептидов, которые определены в блоках с A до I, с использованием порога гестационного возраста при рождении <35 0/7 в сравнении с≥35 0/7 недель.
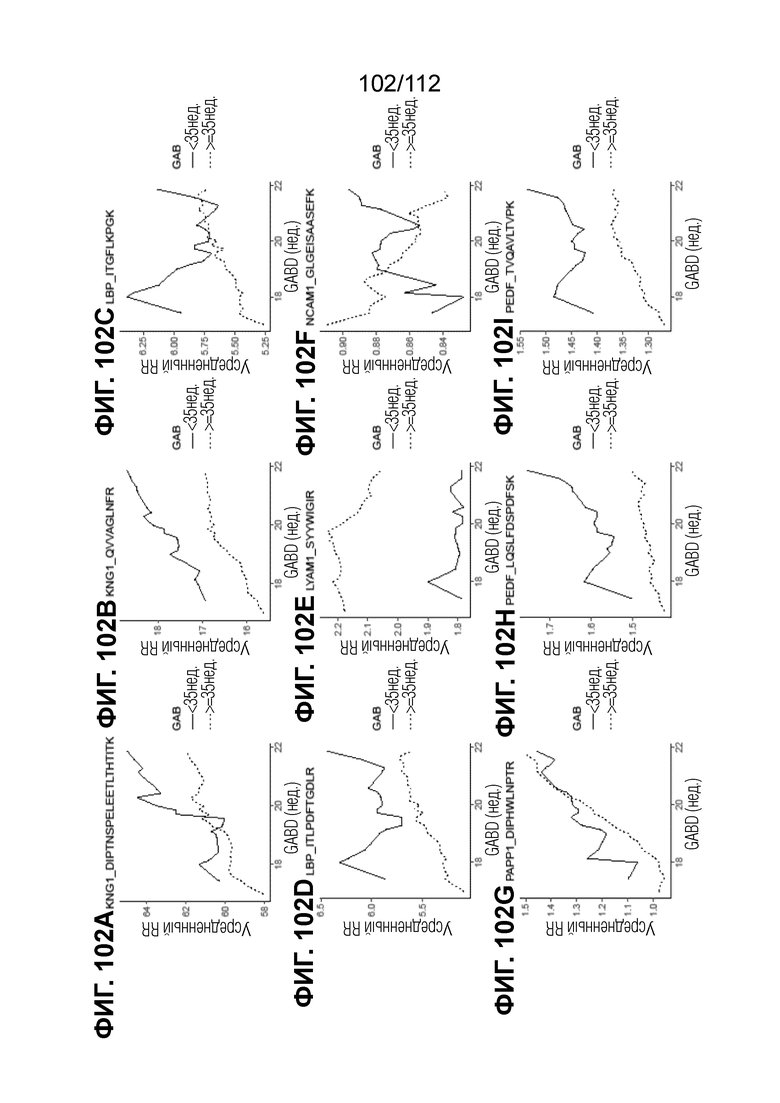
[00146] На фиг. 102 представлены кинетические графики для переходов пептидов, которые определены в блоках с A до I, с использованием порога гестационного возраста при рождении <35 0/7 в сравнении с≥35 0/7 недель.
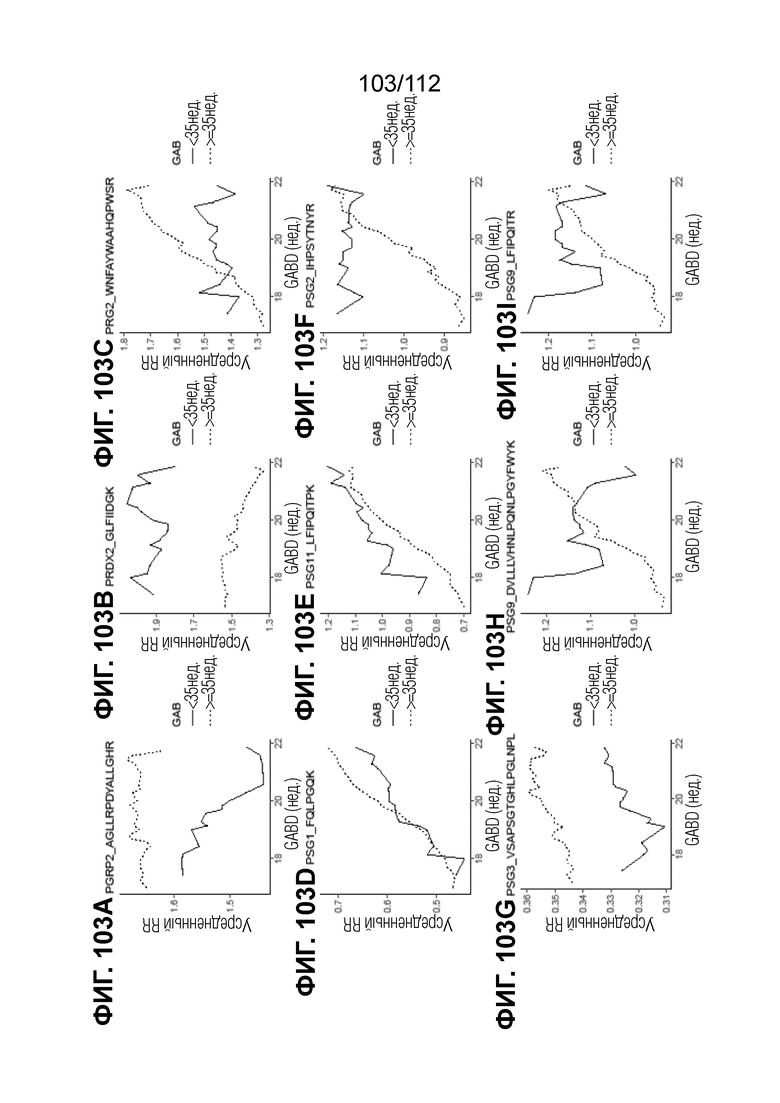
[00147] На фиг. 103 представлены кинетические графики для переходов пептидов, которые определены в блоках с A до I, с использованием порога гестационного возраста при рождении <35 0/7 в сравнении с≥35 0/7 недель.
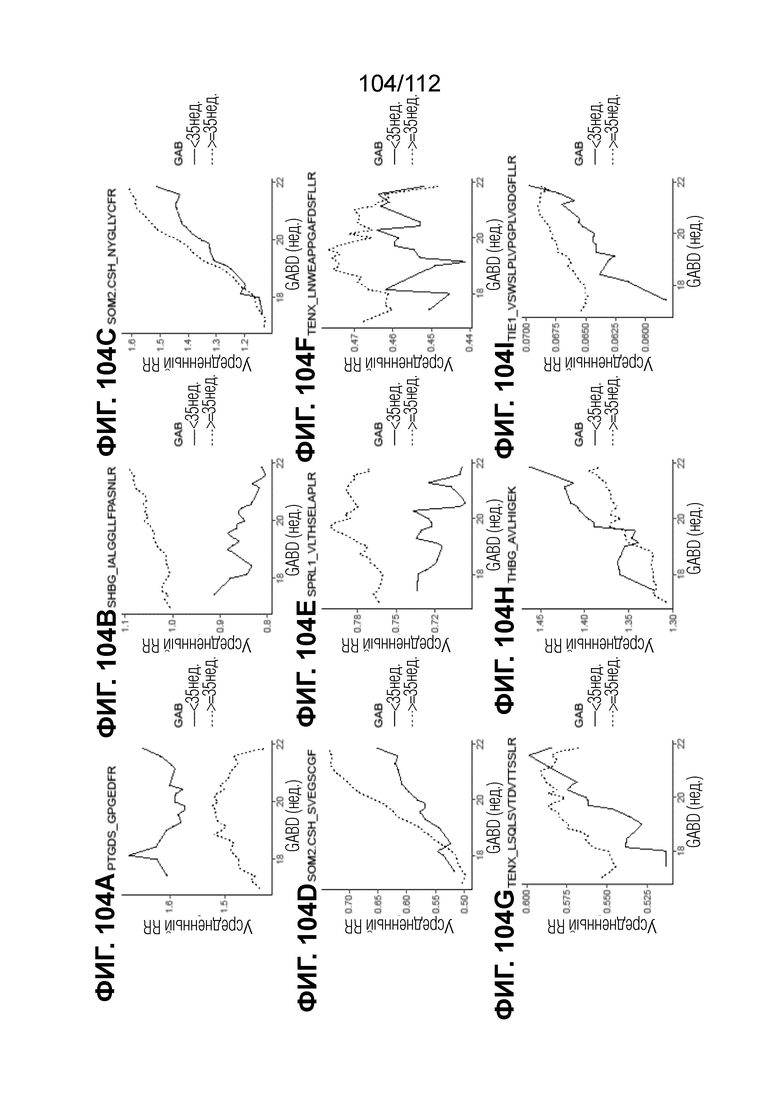
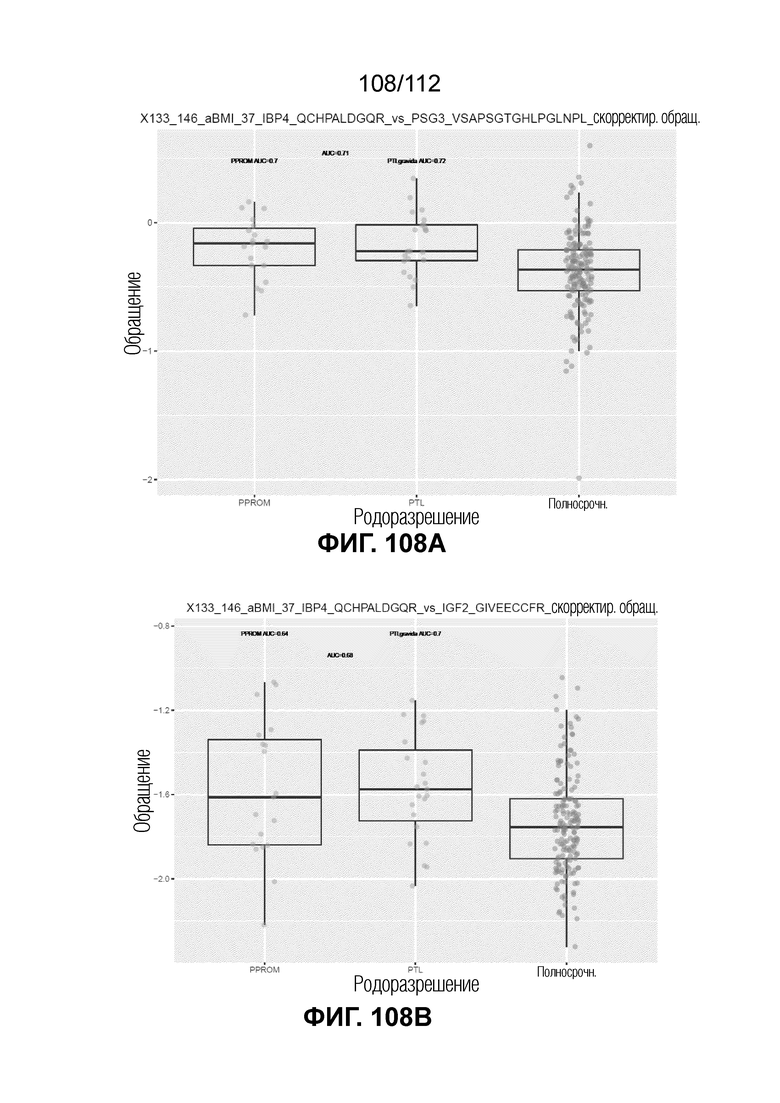
[00148] На фиг. 104 представлены кинетические графики для переходов пептидов, которые определены в блоках с A до I, с использованием порога гестационного возраста при рождении <35 0/7 в сравнении с≥35 0/7 недель.
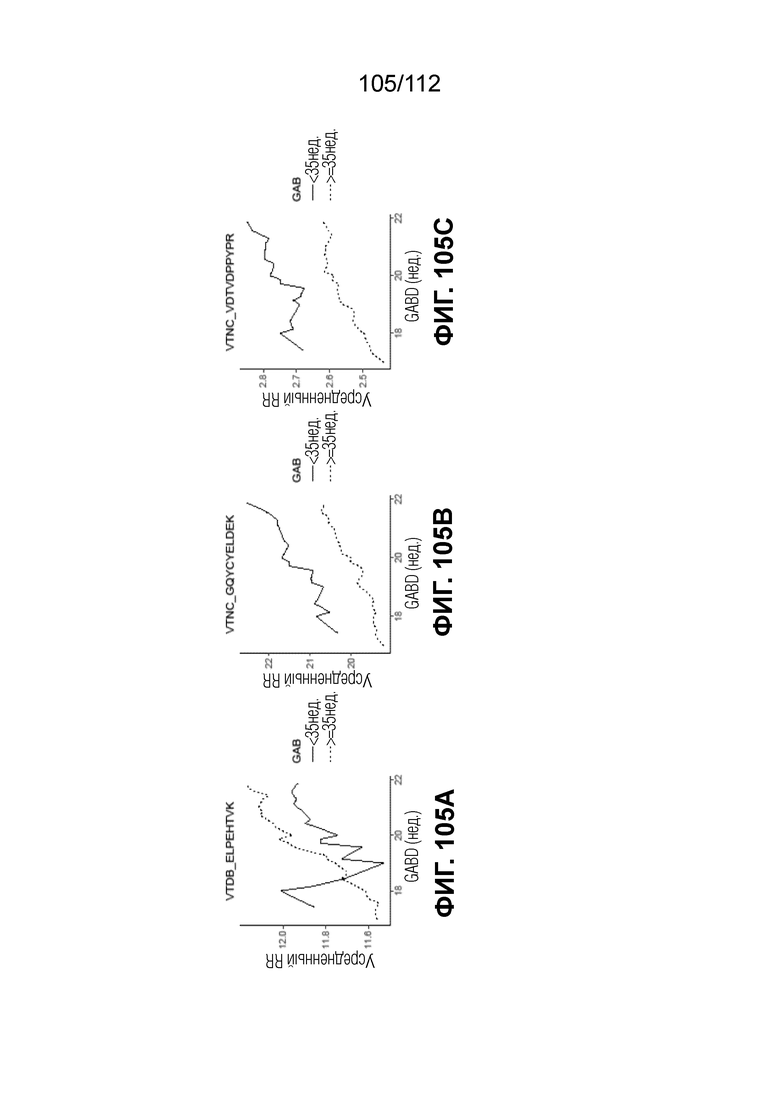
[00149] На фиг. 105 представлены кинетические графики для переходов пептидов, которые определены в блоках с A до C, с использованием порога гестационного возраста при рождении <35 0/7 в сравнении с≥35 0/7 недель.
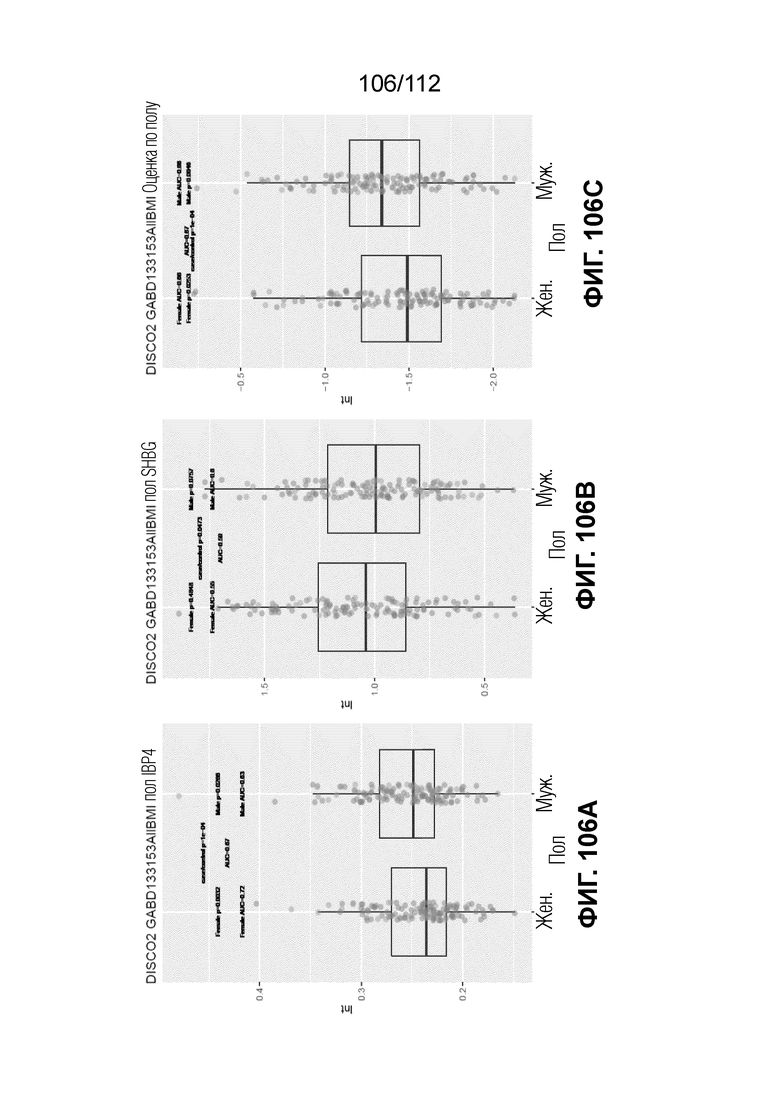
[00150] На фиг. 106 представлены уровни IBP4 и SHBG и значения обращения IBP4/SHBG в случаях sPTB и контролях отдельно.
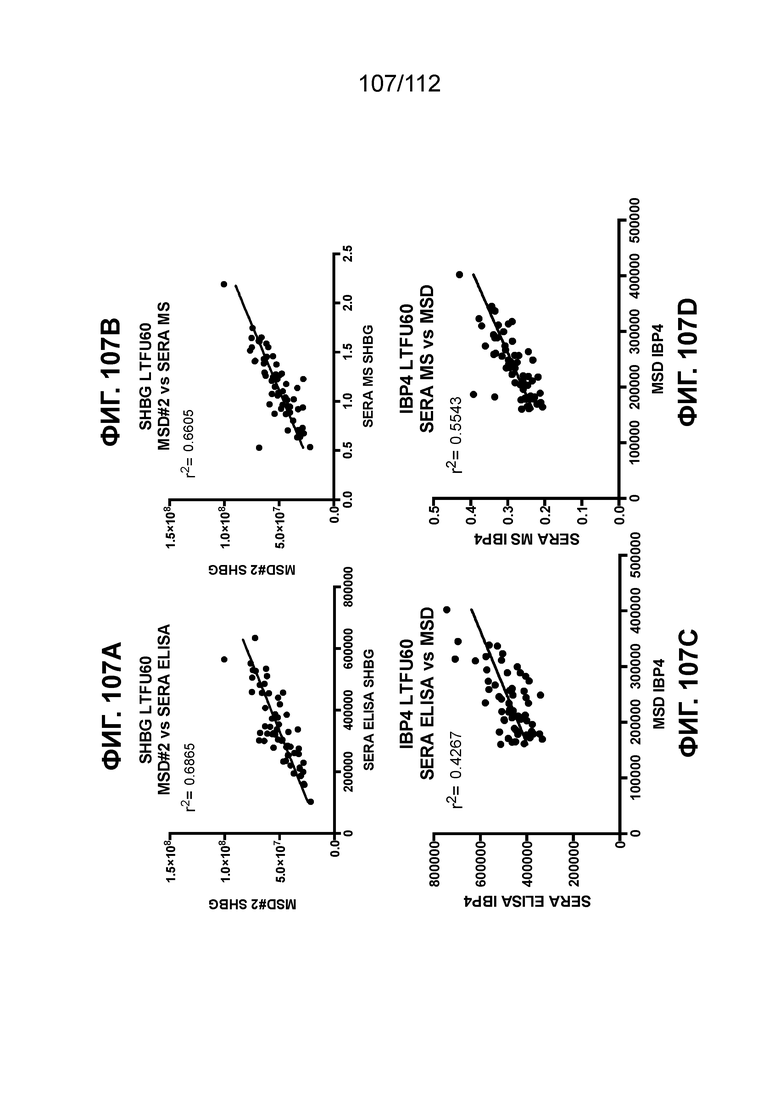
[00151] На фиг. 107 представлена корреляция MSD результатов с использованием коммерческих наборов для ELISA и MS-MRM.
[00152] На фиг. 108 представлены диаграммы размаха, показывающие примеры обращений с хорошей эффективностью в недели 19-20 при преждевременных сокращениях матки в отсутствие PPROM (PTL).
[00153] На фиг. 109 представлены диаграммы размаха, показывающие примеры обращений с хорошей эффективностью в недели 19-20 при преждевременном разрыве околоплодных оболочек (PPROM).
[00154] На фиг. 110 представлена кривая риска, показывающая зависимости между оценкой предиктора (ln IBP4/SHBG) и скорректированным по болезненности относительным риском sPTB (положительное прогностическое значение), используя порог <37 0/7 недель в сравнении с≥37 0/7 недель беременности. Верхняя (пурпурная) линия под графиком кривой риска соответствует sPTB (GAB <35 недель); вторая линия (красная) сверху соответствует sPTB (35≤GAB < 37 недель); третья линия (зеленая) от соответствует TERM (37≤GAB < 39 недель); четвертая линия (синяя) сверху соответствует TERM (39 недель≤GAB).
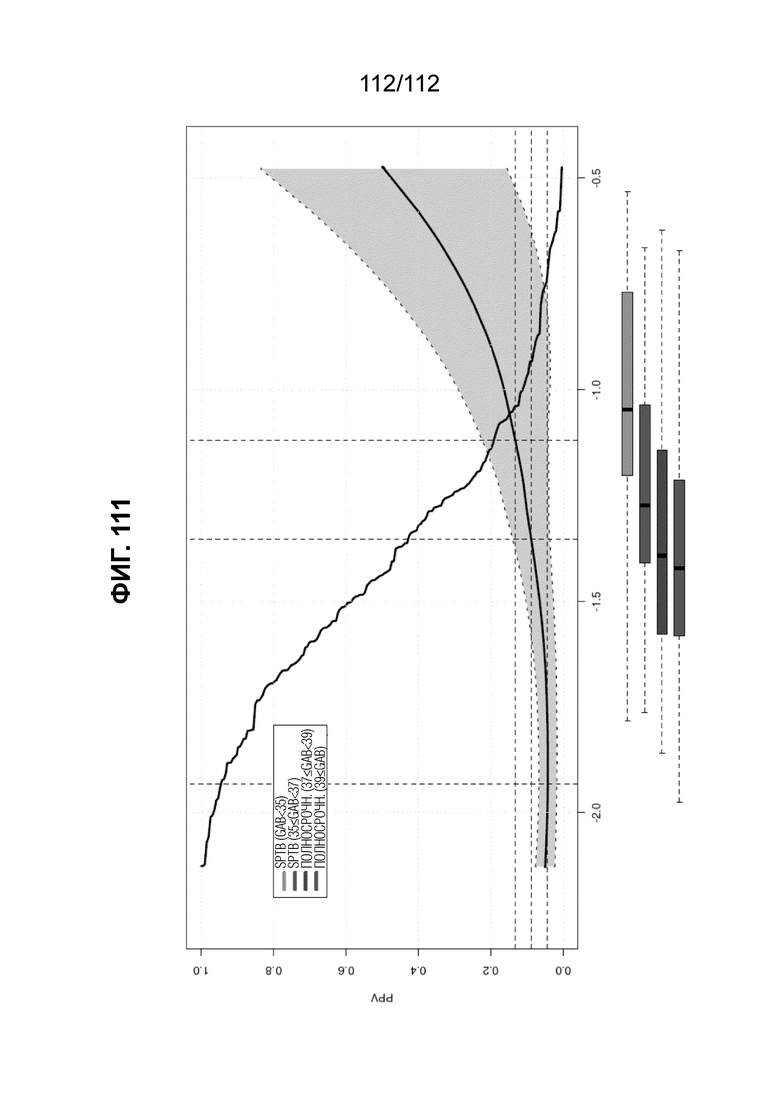
[00155] На фиг. 111 представлена кривая риска, показывающая зависимости между оценкой предиктора (ln IBP4/SHBG) и скорректированным по болезненности относительным риском sPTB (положительное прогностическое значение), используя порог <35 0/7 недель в сравнении с≥35 0/7 недель беременности. Верхняя (пурпурная) линия под графиком кривой риска соответствует sPTB (GAB <35 недель); вторая линия (красная) сверху соответствует sPTB (35≤GAB < 37 недель); третья линия (зеленая) от соответствует TERM (37 < GAB < 39 недель); четвертая линия (синяя) сверху соответствует TERM (39 недель < GAB).
ПОДРОБНОЕ ОПИСАНИЕ
[00156] Настоящее раскрытие основано, в целом, на открытии определенных белков и пептидов в биологических образцах, полученных от беременной женщины, которые дифференциально экспрессированы у беременных женщин, которые имеют повышенный риск преждевременных родов относительно контролей. Настоящее раскрытие, кроме того, в частности, основано, отчасти, на неожиданном открытии того, что значения обращения пар биологических маркеров, описанных в настоящем описании, можно использовать в способах определения вероятности преждевременных родов у беременной женщины с высокой чувствительностью и специфичностью. Белки и пептиды, описанные в настоящем описании в качестве компонентов соотношений и/или пар обращения, служат в качестве биологических маркеров для классификации тестовых образцов, предсказания вероятности преждевременных родов, предсказания вероятности полносрочных родов, предсказания гестационного возраста при рождении (GAB), предсказания времени до родов (TTB) и/или мониторинга прогресса предупредительной терапии у беременной женщины с риском PTB индивидуально, в соотношениях, парах обращения или в панелях биологических маркеров/пар обращения. Значение обращения представляет собой соотношение относительной площади пика биологического маркера под повышающей регуляцией и относительной площади пика биологического маркера под понижающей регуляцией и служит как для того, чтобы нормализовать вариабельность, так и для того, чтобы усиливать диагностический сигнал. Изобретение, отчасти, основано на отборе конкретных биологических маркеров, которые, когда вместе образуют пары, позволяют предсказывать вероятность преждевременных родов на основании значений обращения. Соответственно, в основе настоящего изобретения лежит человеческая изобретательность при отборе конкретных биологических маркеров, которые информативны при образовании пар в новых обращениях.
[00157] Термин «значение обращения» относится к соотношению относительной площади пика анализируемого вещества под повышающей регуляцией и относительной площади пика анализируемого вещества под понижающей регуляцией и служит как для того, чтобы нормализовать вариабельность, так и для того, чтобы усиливать диагностический сигнал. Из всех возможных обращений в узком интервале можно отбирать поднабор на основании индивидуальной одномерной эффективности. Как раскрыто в настоящем описании, соотношение относительной площади пика биологического маркера под повышающей регуляцией и относительной площади пика биологического маркера под понижающей регуляцией, обозначаемое в настоящем описании как значение обращения, можно использовать для того, чтобы идентифицировать надежные и точные классификаторы и предсказывать вероятность преждевременных родов, предсказывать вероятность полносрочных родов, предсказывать гестационный возраст при рождении (GAB), предсказывать время до родов и/или осуществлять мониторинг прогресса предупредительной терапии у беременной женщины. Настоящее изобретение, таким образом, отчасти, основано на идентификации пар биологических маркеров, где относительная экспрессия пары биологических маркеров обращена, что демонстрирует изменение значения обращения между PTB и не PTB. Использование соотношения биологических маркеров в способах, описанных в настоящем описании, корректирует вариабельность, которая является результатом человеческих манипуляций после изъятия биологического образца у беременной женщины. Такую вариабельность можно вводить, например, во время сбора, обработки, истощения, расщепления образца или на какой-либо другой стадии способов, используемых для того, чтобы измерять биологические маркеры, присутствующие в образце, и она не зависит от того, как биологические маркеры ведут себя в природе. Соответственно, изобретение в целом охватывает использование пары обращения в способе диагностирования или прогностирования для того, чтобы снижать вариабельность и/или усиливать, нормализовать или очищать диагностический сигнал.
[00158] Хотя термин значение обращения относится к соотношению относительной площади пика анализируемого вещества под повышающей регуляцией и относительной площади пика анализируемого вещества под понижающей регуляцией и служит как для нормализации вариабельности, так и для усиления диагностического сигнала, также предусмотрено, что пару биологических маркеров по изобретению можно измерять с помощью какого-либо другого средства, например, посредством вычитания, прибавления или умножения относительных площадей пиков. Способы, описанные в настоящем описании, охватывают измерение пар биологических маркеров с помощью таких других средств.
[00159] Этот способ является благоприятным, поскольку он предоставляет самый простой возможный классификатор, который не зависит от данных нормализации, помогает избегать чрезмерной подгонки и дает очень простой экспериментальный тест, который легко реализовать в клинике. Использование пар маркеров на основании изменений значений обращения, которые не зависимы от нормализации данных, сделало возможной разработку клинически релевантных биологических маркеров, описанных в настоящем описании. Поскольку количественное определение какого-либо одного белка подвержено неопределенностям, обусловленным вариабельностью измерений, нормальными флуктуациями и вариациями базового уровня экспрессии у индивидуума, идентификация пар маркеров, которые могут находиться под координированной системной регуляцией, делает возможными надежные способы индивидуализированного диагностирования и прогнозирования.
[00160] Раскрытие предусматривает пары обращения биологических маркеров и связанные панели пар обращения, способы и наборы для определения вероятности преждевременных родов у беременной женщины. Одно основное преимущество по настоящему раскрытию состоит в том, что риск развития преждевременных родов можно оценивать рано во время беременности с тем, чтобы можно было инициировать подходящий мониторинг и клиническое ведение для предотвращения преждевременного родоразрешения своевременным образом. Настоящее изобретение имеет конкретную пользу для женщин, не имеющих каких-либо факторов риска преждевременных родов, которые иначе не будут идентифицированы и не будут получать лечение.
[00161] В качестве примера настоящее раскрытие включает способы создания результата, который можно использовать при определении вероятности преждевременных родов у беременной женщины, посредством получения массива данных, связанных с образцом, где массив данных по меньшей мере содержит количественные данные об относительной экспрессии пар биологических маркеров, которые идентифицированы в качестве демонстрирующих изменения значения обращения, предсказывающего преждевременные роды, и ввода массива данных в аналитический процесс, в котором массив данных используют для того, чтобы создавать результат, который можно использовать при определении вероятности преждевременных родов у беременной женщины. Как дополнительно описано далее, количественные данные могут включать аминокислоты, пептиды, полипептиды, белки, нуклеотиды, нуклеиновые кислоты, нуклеозиды, сахара, жирные кислоты, стероиды, метаболиты, углеводы, липиды, гормоны, антитела, области, представляющие интерес, которые служат в качестве суррогатов биологических макромолекул, и их сочетания.
[00162] В дополнение к конкретным биологическим маркерам, идентифицированным в этом раскрытии, например, по номеру доступа в публичной базе данных, последовательности или ссылке, изобретение также предусматривает использование вариантов биологических маркеров, которые по меньшей мере на 90% или по меньшей мере на 95% или по меньшей мере на 97% идентичны последовательностям, приведенным в качестве примера, и которые известны сейчас или будут открыты позже и которые обладают полезностью для способов по изобретению. Эти варианты могут представлять полиморфизмы, варианты сплайсинга, мутации и т. п. В связи с этим, в данном описании раскрыто множество белков, известных в данной области, в контексте изобретения и предоставлены образцовые номера доступа, связанные с одной или несколькими публичными базами данных, а также образцовые ссылки на статьи, опубликованные в журналах, относящиеся к этим белкам, известным в данной области. Однако специалисты в данной области примут во внимание, что можно легко идентифицировать дополнительные номера доступа и статьи в журналах, которые могут предоставлять дополнительные характеристики раскрытых биологических маркеров, а также что ссылки, приведенные в качестве примера, никак не ограничивают раскрытые биологические маркеры. Как раскрыто в настоящем описании, различные способы и реактивы находят использование в способах по настоящему изобретению. Подходящие образцы в контексте по настоящему изобретению включают, например, кровь, плазму, сыворотку, амниотическую жидкость, вагинальные выделения, слюну и мочу. В некоторых вариантах осуществления биологический образец выбирают из группы, состоящей из цельной крови, плазмы и сыворотки. В конкретном варианте осуществления биологический образец представляет собой сыворотку. Как раскрыто в настоящем описании, биологические маркеры можно обнаруживать с помощью различных анализов и способов, известных в данной области. Как дополнительно описано в настоящем описании, такие анализы включают, без ограничения, анализы на основе масс-спектрометрии (MS), анализы на основе антител, а также анализы, объединяющие аспекты этих двух.
[00163] Белковые биологические маркеры, которые являются компонентами пар обращения, описанных в настоящем описании, включают, например, связывающий инсулиноподобный фактор роста белок 4 (IBP4), связывающий половые гормоны глобулин (SHBG), витронектин (VTNC), группоспецифический компонент (связывающий витамин D белок) (VTDB), катепсин D (лизосомальная аспартилпротеаза) (CATD), специфический β-1-гликопротеин 2 беременности (PSG2), семейство тяжелой цепи интер-альфа-ингибитора трипсина, элемент 4 (ITIH4), L1-подобную молекулу клеточной адгезии (CHL1), компонент комплемента 1, субкомпонент Q, цепь B (C1QB), фибулин 3 (FBLN3), гемопексин (HEMO или HPX), связывающий инсулиноподобный фактор роста белок 6 (IBP6), синтазу простагландина D2 21 кДа (PTGDS).
[00164] В некоторых вариантах осуществления изобретение относится к способу определения вероятности преждевременных родов у беременной женщины, способ включает измерение в биологическом образце, полученном от беременной женщины, значения обращения для по меньшей мере одной пары биологических маркеров, выбранной из группы, содержащей те пары, которые перечислены в какой-либо из сопроводительных фиг. и таблиц, включая фиг. 1.
[00165] В некоторых вариантах осуществления изобретение относится к способу определения вероятности преждевременных родов у беременной женщины, способ включает измерение в биологическом образце, полученном от беременной женщины, значения обращения для по меньшей мере одной пары биологических маркеров, выбранной из группы, состоящей из IBP4/SHBG, VTNC/VTDB, VTNC/SHBG, CATD/SHBG, PSG2/ITIH4, CHL1/ITIH4, PSG2/ITIH4, CHL1/ITIH4, PSG2/C1QB, PSG2/FBLN3, HPX/IBP4, HPX/PTGDS, чтобы определять вероятность преждевременных родов у указанной беременной женщины.
[00166] Изобретение относится к выделенным биологическим маркерам, выбранным из группы, приведенной в таблице 26. Биологические маркеры по изобретению позволяют предсказывать риск преждевременных родов у беременной женщины. В некоторых вариантах осуществления выделенные биологические маркеры выбирают из группы, состоящей из IBP4, SHBG, VTNC, VTDB, CATD, PSG2, ITIH4, CHL1, C1QB, FBLN3, HPX и PTGDS. В некоторых вариантах осуществления выделенные биологические маркеры выбирают из группы, состоящей из IBP4, SHBG, PSG3, LYAM1, IGF2, CLUS, IBP3, INHBC, PSG2, PEDF, CD14 и APOC3.
[00167] Изобретение относится к суррогатным пептидам выделенных биологических маркеров, выбранных из группы, приведенной в таблице 26. В некоторых вариантах осуществления суррогатные пептиды выделенных биологических маркеров выбирают из группы суррогатных пептидов, приведенных в таблице 26. Биологические маркеры по изобретению и их суррогатные пептиды можно использовать в способах предсказания риска преждевременных родов у беременной женщины. В некоторых вариантах осуществления суррогатные пептиды соответствуют выделенным биологическим маркерам, выбранным из группы, состоящей из IBP4, SHBG, VTNC, VTDB,, CATD, PSG2, ITIH4, CHL1, C1QB, FBLN3, HPX и PTGDS. В некоторых вариантах осуществления суррогатные пептиды соответствуют выделенным биологическим маркерам, выбранным из группы, состоящей из IBP4, SHBG, PSG3, LYAM1, IGF2, CLUS, IBP3, INHBC, PSG2, PEDF, CD14 и APOC3.
[00168] Изобретение относится к меченным стабильными изотопами стандартным пептидам (SIS пептидам), соответствующим суррогатным пептидам, выбранным из группы, приведенной в таблице 26. Биологические маркеры по изобретению, их суррогатные пептиды и SIS пептиды можно использовать в способах предсказания риска преждевременных родов у беременной женщины. В некоторых вариантах осуществления SIS пептиды соответствуют суррогатным пептидам выделенных биологических маркеров, выбранных из группы, состоящей из IBP4, SHBG, VTNC, VTDB,, CATD, PSG2, ITIH4, CHL1, C1QB, FBLN3, HPX и PTGDS. В некоторых вариантах осуществления SIS пептиды соответствуют суррогатным пептидам выделенных биологических маркеров, выбранных из группы, состоящей из IBP4, SHBG, PSG3, LYAM1, IGF2, CLUS, IBP3, INHBC, PSG2, PEDF, CD14 и APOC3.
[00169] В некоторых вариантах осуществления изобретение относится к паре выделенных биологических маркеров IBP4/SHBG, где пара биологических маркеров демонстрирует изменение значения обращения между беременными женщинами с риском преждевременных родов по сравнению с полносрочными контролями. В дополнительном варианте осуществления изобретение относится к паре выделенных биологических маркеров IBP4/SHBG, где пара биологических маркеров демонстрирует более высокое соотношение у беременных женщин с риском преждевременных родов по сравнению с полносрочными контролями.
[00170] В некоторых вариантах осуществления изобретение относится к способу определения вероятности преждевременных родов у беременной женщины, способ включает измерение в биологическом образце, полученном от беременной женщины, значения обращения для по меньшей мере одной пары биологических маркеров, выбранной из группы, состоящей из IBP4/SHBG, VTNC/VTDB, VTNC/SHBG и CATD/SHBG, чтобы определять вероятность преждевременных родов у указанной беременной женщины. В дополнительных вариантах осуществления образец получают между 19 и 21 неделями GABD. В дополнительных вариантах осуществления образец получают между 19 и 22 неделями GABD.
[00171] В некоторых вариантах осуществления изобретение относится к способу определения вероятности преждевременных родов у беременной женщины, способ включает измерение в биологическом образце, полученном от беременной женщины, значения обращения для IBP4/SHBG, чтобы определять вероятность преждевременных родов у указанной беременной женщины. В дополнительных вариантах осуществления образец получают между 19 и 21 неделями GABD. В дополнительных вариантах осуществления образец получают между 19 и 22 неделями GABD.
[00172] В некоторых вариантах осуществления изобретение относится к способу определения вероятности преждевременных родов у беременной женщины, способ включает измерение в биологическом образце, полученном от беременной женщины, значения обращения для по меньшей мере одной пары биологических маркеров, выбранной из группы, состоящей из IBP4/SHBG, VTNC/VTDB, VTNC/SHBG, CATD/SHBG, PSG2/ITIH4, CHL1/ITIH4, PSG2/C1QB, PSG2/FBLN3, HPX/IBP4, HPX/PTGDS, чтобы определять вероятность преждевременных родов у указанной беременной женщины, где существование изменения значения обращения между беременной женщиной и полносрочным контролем определяет вероятность преждевременных родов у беременной женщины. В дополнительных вариантах осуществления образец получают между 19 и 21 неделями GABD. В дополнительных вариантах осуществления образец получают между 19 и 22 неделями GABD.
[00173] В варианты осуществления изобретения включены итерационные способы определения вероятности преждевременных родов у беременной женщины, способ включает измерение в биологическом образце, полученном от беременной женщины, значения обращения для по меньшей мере одной пары биологических маркеров, выбранной из группы, состоящей из IBP4/SHBG, VTNC/VTDB, VTNC/SHBG, CATD/SHBG, PSG2/ITIH4, CHL1/ITIH4, PSG2/C1QB, PSG2/FBLN3, HPX/IBP4, HPX/PTGDS и какой-либо другой пары биологических маркеров, выбранных из белков, описанных и/или приведенных в качестве примера в настоящем описании, чтобы определять вероятность преждевременных родов у указанной беременной женщины, где существование изменения значения обращения между беременной женщиной и полносрочным контролем определяет вероятность преждевременных родов у беременной женщины. Итерационная эффективность способов, описанных в настоящем описании, включает последующие измерения, получаемые из одного образца, а также получение последующих образцов для измерения. Например, если определяют, что вероятность преждевременных родов у беременной женщины, которую можно выражать в виде оценки риска, выше конкретного значения, способ можно повторять с использованием отличающейся пары обращения из того же образца или той же или отличающейся пары обращения из последующего образца, чтобы дополнительно стратифицировать риск sPTB.
[00174] В дополнение к конкретным биологическим маркерам раскрытие, кроме того, включает варианты биологических маркеров, которые приблизительно на 90%, приблизительно на 95% или приблизительно на 97% идентичны образцовым последовательностям. Варианты, как используют в настоящем описании, включают полиморфизмы, варианты сплайсинга, мутации и т. п. Несмотря на то, что описание приведено со ссылкой на белковые биологические маркеры, изменения значения обращения можно идентифицировать в уровнях белков или экспрессии генов для пар биологических маркеров.
[00175] Дополнительные маркеры можно выбирать из одного или нескольких признаков риска, включая в качестве неограничивающих примеров материнские характеристики, медицинский анамнез, анамнез прошлой беременности и акушерский анамнез. Такие дополнительные маркеры могут включать, например, предыдущую низкую массу при рождении или преждевременное родоразрешение, множественные самопроизвольные аборты 2-го триместра, предшествующий вызванный аборт первого триместра, семейные факторы и факторы различных поколений, анамнез бесплодия, неспособность к деторождению, аномалии плаценты, аномалии матки и шейки матки, измерения малой длины шейки матки, кровотечения при беременности, задержку внутриутробного развития, экспозицию диэтилстильбэстрола in utero, множественные беременности, пол младенца, небольшой рост, низкую массу перед беременностью, низкий или высокий индекс массы тела, диабет, гипертензию, урогенитальные инфекции (т. е. инфекции мочевых путей), астму, беспокойство и депрессию, астму, гипертензию, гипотиреоидизм. Демографические признаки риска для преждевременных родов могут включать, например, возраст матери, расовую/этническую принадлежность, незамужнее положение, низкий социально-экономический статус, возраст матери, физическую активность связанную с трудовой деятельностью, профессиональные воздействия и экологические воздействия, а также стресс. Дополнительные признаки риска могут включать недостаточное наблюдение за беременностью, курение сигарет, использование марихуаны и других запрещенных средств, использование кокаина, употребление алкоголя, прием кофеина, увеличение массы матери, пищевой рацион, половую активность во время поздней беременности и физические активности в свободное время (Preterm Birth: Causes, Consequences, and Prevention, Institute of Medicine (US) Committee on Understanding Premature Birth and Assuring Healthy Outcomes; ред. Behrman RE, Butler AS, Washington (DC): National Academies Press (US); 2007). Дополнительные признаки риска, которые можно использовать в качестве маркеров, можно идентифицировать с использованием алгоритмов обучения, известных в данной области, таких как линейный дискриминантный анализ, классификация способом опорных векторов, рекурсивная элиминация признаков, прогностический анализ микрочипа, логистическая регрессия, CART, FlexTree, LART, случайный лес, MART и/или регрессионный анализ выживаемости, которые известны специалистам в данной области и дополнительно описаны в настоящем описании.
[00176] Следует отметить, что, как используют в этом описании и приложенной формуле изобретения, формы единственного числа включают множественное число до тех пор, пока содержание явно не диктует иное. Таким образом, например, упоминание о «биологическом маркере» включает смесь двух или больше биологических маркеров и т. п.
[00177] Термин «приблизительно», в частности, по отношению к приведенному количеству, значит, что включены отклонения±5%.
[00178] Как используют в этой заявке, включая приложенную формулу изобретения, формы единственного числа включают множественное число до тех пор, пока содержание явно не диктует иное, и используют взаимозаменяемо с «по меньшей мере один» и «один или несколько».
[00179] Как используют в настоящем описании, термины «содержит», «содержащий», «включает», «включающий» и какие-либо их вариации предназначены для того, чтобы покрывать неисключительное включение, так что процесс, способ, продукт, характеризуемый способом его получения, или композиция вещества, которая содержит или включает элемент или список элементов, не содержит только эти элементы, но может включать другие элементы, в явной форме не перечисленные или свойственные для такого процесса, способа, продукта, характеризуемого способом его получения, или композиции вещества.
[00180] Как используют в настоящем описании, термин «панель» относится к композиции, такой как массив или совокупность, которая содержит один или несколько биологических маркеров. Термин также может относиться к профилю или индексу профилей экспрессии одного или нескольких биологических маркеров, описанных в настоящем описании. Число биологических маркеров, используемых для панели биологических маркеров, основано на значении чувствительности и специфичности для конкретной комбинации значений биологических маркеров.
[00181] Как используют в настоящем описании, и если не указано иное, термины «выделенный» и «очищенный» в целом описывают композицию вещества, которая удалена из ее нативной среды (например, природной среды, если она встречается в природе) и, таким образом, изменена руками человека относительно ее природного состояния с тем, чтобы обладать заметно отличающимися характеристиками в отношению по меньшей мере одного из структуры, функции и свойств. Выделенный белок или нуклеиновая кислота отличается от того, как она существует в природе, и включает синтетические пептиды и белки.
[00182] Термин «биологический маркер» относится к биологической молекуле или фрагменту биологической молекулы, изменение и/или обнаружение которой может коррелировать с конкретным физическим состоянием. Термины «маркер» и «биологический маркер», используют взаимозаменяемо на всем протяжении раскрытия, например, биологические маркеры по настоящему изобретению коррелирует с повышенной вероятностью преждевременных родов. Такие биологические маркеры включают любое подходящее анализируемое вещество, но не ограничиваясь этим, биологические молекулы, содержащие нуклеотиды, нуклеиновые кислоты, нуклеозиды, аминокислоты, сахара, жирные кислоты, стероиды, метаболиты, пептиды, полипептиды, белки, углеводы, липиды, гормоны, антитела, области, представляющие интерес, которые служат в качестве суррогатов для биологических макромолекул, и их сочетания (например, гликопротеины, рибонуклеопротеины, липопротеины). Термин также охватывает части или фрагменты биологической молекулы, например, пептидный фрагмент белка или полипептид, который содержит по меньшей мере 5 последовательных аминокислотных остатков, по меньшей мере 6 последовательных аминокислотных остатков, по меньшей мере 7 последовательных аминокислотных остатков, по меньшей мере 8 последовательных аминокислотных остатков, по меньшей мере 9 последовательных аминокислотных остатков, по меньшей мере 10 последовательных аминокислотных остатков, по меньшей мере 11 последовательных аминокислотных остатков, по меньшей мере 12 последовательных аминокислотных остатков, по меньшей мере 13 последовательных аминокислотных остатков, по меньшей мере 14 последовательных аминокислотных остатков, по меньшей мере 15 последовательных аминокислотных остатков, по меньшей мере 5 последовательных аминокислотных остатков, по меньшей мере 16 последовательных аминокислотных остатков, по меньшей мере 17 последовательных аминокислотных остатков, по меньшей мере 18 последовательных аминокислотных остатков, по меньшей мере 19 последовательных аминокислотных остатков, по меньшей мере 20 последовательных аминокислотных остатков, по меньшей мере 21 последовательный аминокислотный остаток, по меньшей мере 22 последовательных аминокислотных остатка, по меньшей мере 23 последовательных аминокислотных остатка, по меньшей мере 24 последовательных аминокислотных остатка, по меньшей мере 25 последовательных аминокислотных остатков или больше последовательных аминокислотных остатков.
[00183] Как используют в настоящем описании, термин «суррогатный пептид» относится к пептиду, который выбирают для того, чтобы он служил в качестве суррогата для количественного определения биологического маркера, представляющего интерес, в MRM конфигурации анализа. Количественное определение суррогатных пептидов лучше всего осуществляют с использованием меченных стабильными изотопами стандартных суррогатных пептидов («SIS суррогатных пептидов» или «SIS пептидов») в сочетании с MRM способом обнаружения. Суррогатный пептид может быть синтетическим. SIS суррогатный пептид можно синтезировать интенсивно меченным например, аргинином или лизином или какой-либо другой аминокислотой на C-конце пептида, чтобы он служил в качестве внутреннего стандарта в MRM анализе. SIS суррогатный пептид не является встречающимся в природе пептидом и обладает заметно отличающимися структурой и свойствами по сравнению с его встречающимся в природе эквивалентом.
[00184] В некоторых вариантах осуществления изобретение относится к способу определения вероятности преждевременных родов у беременной женщины, способ включает измерение в биологическом образце, полученном от беременной женщины, соотношения для по меньшей мере одной пары биологических маркеров, выбранной из группы, состоящей из IBP4/SHBG, VTNC/VTDB, VTNC/SHBG, CATD/SHBG, PSG2/ITIH4, CHL1/ITIH4, PSG2/C1QB, PSG2/FBLN3, HPX/IBP4, HPX/PTGDS, чтобы определять вероятность преждевременных родов у указанной беременной женщины, где существование изменения соотношения между беременной женщиной и полносрочным контролем определяет вероятность преждевременных родов у беременной женщины. В некоторых вариантах осуществления соотношение может включать белок под повышающей регуляцией в числителе, белок под понижающей регуляцией в знаменателе или оба. Например, как показано на примере в настоящем описании, IBP4/SHBG представляет собой соотношение белка под повышающей регуляцией в числителе и белка под понижающей регуляцией в знаменателе, которое определяют в настоящем описании как «обращение». В ситуациях, когда соотношение включает белок под повышающей регуляцией в числителе или белок под понижающей регуляцией в знаменателе, не регулируемый белок будет служить для нормализации (например, для снижения преданалитической или аналитической вариабельности). В конкретном случае соотношения, которое представляет собой «обращение», возможны как усиление, так и нормализация. Понятно, что способы по изобретению не ограничены конкретным поднабором обращений, но также охватывают соотношения биологических маркеров.
[00185] Как используют в настоящем описании, термин «обращение» относится к соотношению измеряемого значения анализируемого вещества под повышающей регуляцией и значения анализируемого вещества под понижающей регуляцией. В некоторых вариантах осуществления само значение анализируемого вещества представляет собой соотношение площади пика эндогенного анализируемого вещества и площади пика соответствующего стабильного изотопного стандартного анализируемого вещества, упоминаемого в настоящем описании как: коэффициент ответа или удельное соотношение.
[00186] Как используют в настоящем описании, термин «пара обращения» относится к биологическим маркерам в парах, которые демонстрируют изменение значения между классами, подлежащими сравнению. Обнаружение обращений в концентрациях белков или уровнях экспрессии генов устраняет необходимость нормализации данных или установления порогов в рамках всей популяции. В некоторых вариантах осуществления пара обращения представляет собой пару выделенных биологических маркеров IBP4/SHBG, где пара обращения демонстрирует изменение значения обращения между беременными женщинами с риском преждевременных родов по сравнению с полносрочными контролями. В дополнительном варианте осуществления пара обращения IBP4/SHBG демонстрирует более высокое соотношение у беременных женщин с риском преждевременных родов по сравнению с полносрочными контролями. Определение какой-либо пары обращения охватывает соответствующую пару обращения, в которой индивидуальные биологические маркеры поменяны между числителем и знаменателем. Специалист в данной области примет во внимание, что такая соответствующая пара обращения в равной мере информативна в отношении ее предсказательной мощности.
[00187] Термин «значение обращения» относится к соотношению относительной площади пика анализируемого вещества под повышающей регуляцией и относительной площади пика анализируемого вещества под понижающей регуляцией и служит как для нормализации вариабельности, так и для усиления диагностического сигнала. Из всех возможных обращений с узким интервалом можно отбирать поднабор на основании индивидуальной одномерной эффективности. Как раскрыто в настоящем описании, соотношение относительной площади пика биологического маркера под повышающей регуляцией и относительной площади пика биологического маркера под понижающей регуляцией, упоминаемое в настоящем описании как значение обращения, можно использовать для того, чтобы идентифицировать надежные и точные классификаторы и предсказывать вероятность преждевременных родов, предсказывать вероятность полносрочных родов, предсказывать гестационный возраст при рождении (GAB), предсказывать время до родов и/или осуществлять мониторинг прогресса предупредительной терапии у беременной женщины.
[00188] Этот способ с обращением является благоприятным, поскольку он предоставляет самый простой возможный классификатор, который не зависит от нормализации данных, помогает избегать чрезмерной подгонки и ведет к очень простому экспериментальному тесту, который легко реализовать в клинике. Использование пар биологических маркеров на основании обращений, которые не зависят от нормализации данных, как раскрыто в настоящем описании, имеет огромную мощность в качестве способа идентификации клинически релевантных биологических маркеров PTB. Поскольку количественное определение какого-либо одного белка подвержено неопределенностям, обусловленным вариабельностью измерения, нормальными флуктуациями и вариациями базового уровня экспрессии у индивидуума, идентификация пар маркеров, которые могут находиться под координированной системной регуляцией, должна оказываться более надежной для индивидуализированного диагностирования и прогнозирования.
[00189] Изобретение относится к композиции, содержащей пару выделенных биологических маркеров, выбранную из группы, состоящей из IBP4/SHBG, VTNC/VTDB, VTNC/SHBG, CATD/SHBG, PSG2/ITIH4, CHL1/ITIH4, PSG2/C1QB, PSG2/FBLN3, HPX/IBP4 и HPX/PTGDS, где пара биологических маркеров демонстрирует изменение значения обращения между беременными женщинами с риском преждевременных родов и полносрочными контролями. В одном из вариантов осуществления композиции содержат меченные стабильными изотопами стандартные пептиды (SIS пептиды) для суррогатных пептидов, полученных из каждого из указанных биологических маркеров.
[00190] В конкретных вариантах осуществления изобретение относится к паре выделенных биологических маркеров, состоящей из IBP4 и SHBG, где пара демонстрирует изменение значения обращения между беременными женщинами с риском преждевременных родов и полносрочными контролями.
[00191] IBP4 является членом семейства связывающих инсулиноподобный фактор роста белков (IBP), которые осуществляют отрицательную регуляцию инсулиноподобных факторов роста IGF1 и IGF2. (Insulin-like growth factor I and II regulate the life cycle of trophoblast in the developing human placenta. Am J Physiol, Cell Physiol. 2008;294(6):C1313-22). IBP4 экспрессируют с помощью синцитиотрофобластов (Crosley et al., IGFBP-4 and -5 are expressed in first-trimester villi and differentially regulate the migration of HTR-8/SVneo cells. Reprod Biol Endocrinol. 2014;12(1):123), и он является преобладающим IBP, экспрессируемым вневорсинчатыми трофобластами (Qiu et al. Significance of IGFBP-4 in the development of fetal growth restriction. J Clin Endocrinol Metab. 2012;97(8):E1429-39). По сравнению с полносрочными беременностями, материнские уровни IBP4 во время ранней беременности выше при беременностях, осложненных задержкой развития плода и предэклампсией. (Qiu et al., выше, 2012)
[00192] SHBG регулирует доступность биологически активных несвязанных стероидных гормонов. Hammond GL. Diverse roles for sex hormone-binding globulin in reproduction. Biol Reprod. 2011;85(3):431-41. Уровни SHBG в плазме возрастают в 5-10 раз во время беременности (Anderson DC. Sex-hormone-binding globulin. Clin Endocrinol (Oxf). 1974;3(1):69-96), и существуют свидетельства внепеченочной экспрессии, включая плацентарные трофобластные клетки. (Larrea et al. Evidence that human placenta is a site of sex hormone-binding globulin gene expression. J Steroid Biochem Mol Biol. 1993;46(4):497-505) Физиологически уровни SHBG отрицательно коррелируют с уровнями триглицеридов, инсулина и BMI. (Simó et al. Novel insights in SHBG regulation and clinical implications. Trends Endocrinol Metab. 2015;26(7):376-83) Эффект BMI, оказываемый на уровни SHBG, может отчасти объяснять повышенную предсказательну5 эффективность при BMI-стратификации.
[00193] Интраамниотическая инфекция и воспаление связаны с PTB, поскольку имеют место повышенные уровни провоспалительных цитокинов, включая TNF-α и IL1-β. (Mendelson CR. Minireview: fetal-maternal hormonal signaling in pregnancy and labor. Mol Endocrinol. 2009;23(7):947-54; Gomez-Lopez et al. Immune cells in term and preterm labor. Cell Mol Immunol. 2014;11(6):571-81). Транскрипция SHBG в печени подавлена с помощью IL1-β и опосредованной NF-κB передачи сигналов TNF-α (Simó et al. Novel insights in SHBG regulation and clinical implications. Trends Endocrinol Metab. 2015;26(7):376-83), пути, вовлеченного в инициацию нормальных и аномальных родов (Lindström TM, Bennett PR. The role of nuclear factor kappa B in human labour. Reproduction. 2005;130(5):569-81). Более низкие уровни SHBG у женщин, которым грозит sPTB, могут представлять собой результат инфекции и/или воспаления. Таким образом, SHBG может быть критичным для контроля действия андрогенов и эстрогенов в фетоплацентарном комплексе в ответ на нисходящие воспалительные сигналы.
[00194] В одном из вариантов осуществления изобретение относится к композиции, содержащей пару суррогатных пептидов, соответствующую паре биологических маркеров, выбранных из группы, состоящей из IBP4/SHBG, VTNC/VTDB, VTNC/SHBG, CATD/SHBG, PSG2/ITIH4, CHL1/ITIH4, PSG2/C1QB, PSG2/FBLN3, HPX/IBP4 и HPX/PTGDS, где пара биологических маркеров демонстрирует изменение значения обращения между беременными женщинами с риском преждевременных родов и полносрочными контролями.
[00195] В дополнительном варианте осуществления изобретение относится к панели из по меньшей мере двух пар биологических маркеров, выбранных из группы, состоящей из IBP4/SHBG, VTNC/VTDB, VTNC/SHBG, CATD/SHBG, PSG2/ITIH4, CHL1/ITIH4, PSG2/C1QB, PSG2/FBLN3, HPX/IBP4 и HPX/PTGDS, где каждая из пар демонстрирует изменение значения обращения между беременными женщинами с риском преждевременных родов и полносрочными контролями.
[00196] В дополнительном варианте осуществления изобретение относится к панели из по меньшей мере двух пар суррогатных пептидов, каждая пара суррогатных пептидов соответствует паре биологических маркеров, выбранной из группы, состоящей из IBP4/SHBG, VTNC/VTDB, VTNC/SHBG, CATD/SHBG, PSG2/ITIH4, CHL1/ITIH4, PSG2/C1QB, PSG2/FBLN3, HPX/IBP4 и HPX/PTGDS, где каждая из пар демонстрирует изменение значения обращения между беременными женщинами с риском преждевременных родов и полносрочными контролями.
[00197] В одном из вариантов осуществления изобретение относится к способу определения вероятности преждевременных родов у беременной женщины, способ включает измерение в биологическом образце, полученном от беременной женщины, значения обращения для по меньшей мере одной пары биологических маркеров, выбранной из группы, состоящей из IBP4/SHBG, VTNC/VTDB, VTNC/SHBG, CATD/SHBG, PSG2/ITIH4, CHL1/ITIH4, PSG2/C1QB, PSG2/FBLN3, HPX/IBP4 и HPX/PTGDS, чтобы определять вероятность преждевременных родов у беременной женщины.
[00198] В другом варианте осуществления изобретение относится к способу определения вероятности преждевременных родов у беременной женщины, способ включает измерение в биологическом образце, полученном от беременной женщины, изменения значения обращения для панели из по меньшей мере двух пар биологических маркеров, выбранных из группы, состоящей из IBP4/SHBG, VTNC/VTDB, VTNC/SHBG, CATD/SHBG, PSG2/ITIH4, CHL1/ITIH4, PSG2/C1QB, PSG2/FBLN3, HPX/IBP4 и HPX/PTGDS, чтобы определять вероятность преждевременных родов у беременной женщины. В некоторых вариантах осуществления значение обращения раскрывает существование изменения значения обращения между беременной женщиной и полносрочным контролем и отражает вероятность преждевременных родов у беременной женщины. В некоторых вариантах осуществления стадия измерения включает измерение суррогатных пептидов биологических маркеров в биологическом образце, полученном от беременной женщины.
[00199] В одном из вариантов осуществления изобретение относится к способу определения вероятности преждевременных родов у беременной женщины, способ включает измерение в биологическом образце, полученном от беременной женщины, значения обращения для по меньшей мере одной пары биологических маркеров, выбранных из группы, состоящей из биологических маркеров, перечисленных в какой-либо из таблиц с 1 до 77 и на фиг. с 1 до 111, у беременной женщины, чтобы определять вероятность преждевременных родов у беременной женщины.
[00200] В дополнительном варианте осуществления изобретение относится к способу определения вероятности преждевременных родов у беременной женщины, способ включает измерение в биологическом образце, полученном от беременной женщины, значения обращения для по меньшей мере одной пары биологических маркеров, выбранной из группы, состоящей из пар биологических маркеров, точно определенных в таблицах с 27 до 59, с 61 до 72, 76 и 77, у беременной женщины, чтобы определять вероятность преждевременных родов у беременной женщины.
[00201] В дополнительном варианте осуществления, изобретение относится к способу определения вероятности преждевременных родов у беременной женщины, способ включает измерение в биологическом образце, полученном от беременной женщины, значения обращения для по меньшей мере одной пары биологических маркеров, выбранных из группы, состоящей из биологических маркеров, перечисленных в таблице 26, у беременной женщины, чтобы определять вероятность преждевременных родов у беременной женщины.
[00202] В другом варианте осуществления изобретение относится к способу определения вероятности преждевременных родов у беременной женщины, способ включает измерение в биологическом образце, полученном от беременной женщины, изменения значения обращения для панели из по меньшей мере двух пар биологических маркеров, выбранных из группы, состоящей из пар биологических маркеров, точно определенных в какой-либо из таблиц с 1 до 77 и на фиг. с 1 до 111, у беременной женщины, чтобы определять вероятность преждевременных родов у беременной женщины. В некоторых вариантах осуществления значение обращения раскрывает существование изменения значения обращения между беременной женщиной и полносрочным контролем и отражает вероятность преждевременных родов у беременной женщины. В некоторых вариантах осуществления стадия измерения включает измерение суррогатных пептидов биологических маркеров в биологическом образце, полученном от беременной женщины.
[00203] В другом варианте осуществления изобретение относится к способу определения вероятности преждевременных родов у беременной женщины, способ включает измерение в биологическом образце, полученном от беременной женщины, изменения значения обращения для панели из по меньшей мере двух пар биологических маркеров, выбранных из группы, состоящей из пар биологических маркеров, которые точно определены в таблицах с 27 до 59, с 61 до 72, 76 и 77, у беременной женщины, чтобы определять вероятность преждевременных родов у беременной женщины. В некоторых вариантах осуществления значение обращения раскрывает существование изменения значения обращения между беременной женщиной и полносрочным контролем и отражает вероятность преждевременных родов у беременной женщины. В некоторых вариантах осуществления стадия измерения включает измерение суррогатных пептидов биологических маркеров в биологическом образце, полученном от беременной женщины.
[00204] В другом варианте осуществления изобретение относится к способу определения вероятности преждевременных родов у беременной женщины, способ включает измерение в биологическом образце, полученном от беременной женщины, изменения значения обращения для панели из по меньшей мере двух пар биологических маркеров, выбранных из группы, состоящей из биологических маркеров, которые точно определены в таблице 26, у беременной женщины, чтобы определять вероятность преждевременных родов у беременной женщины. В некоторых вариантах осуществления значение обращения раскрывает существование изменения значения обращения между беременной женщиной и полносрочным контролем и отражает вероятность преждевременных родов у беременной женщины. В некоторых вариантах осуществления стадия измерения включает измерение суррогатных пептидов биологических маркеров в биологическом образце, полученном от беременной женщины.
[00205] Для способов, направленных на предсказание времени до родов, понятно, что «роды» обозначают роды после самопроизвольного начала родового акта, с разрывом оболочек или без него.
[00206] Несмотря на то, что описано и иллюстрировано со ссылкой на способы определения вероятности преждевременных родов у беременной женщины, настоящее раскрытие аналогичным образом применимо к способам предсказания гестационного возраста при рождении (GAB), способам предсказания полносрочных родов, способам определения вероятности полносрочных родов у беременной женщины, а также к способам предсказания времени до родов (TTB) у беременной женщины. Специалисту в данной области будет ясно, что каждый из указанных выше способов имеет конкретные и существенные полезности и эффекты в отношении соображений здоровья матери и плода.
[00207] Кроме того, несмотря на то, что описано и иллюстрировано со ссылкой на способы определения вероятности преждевременных родов у беременной женщины, настоящее раскрытие аналогичным образом в применимо к способам предсказания ненормального глюкозного теста, диабета беременных, гипертензии, предэклампсии, задержки внутриутробного развития, мертворождения, задержки развития плода, синдрома HELLP, олигогидрамниона, хориоамнионита, хориоамнионита, предлежания плаценты, приросшей плаценты, отслоения, отрыва плаценты, плацентарной геморрагии, преждевременного разрыва околоплодных оболочек, преждевременных сокращений матки, неудовлетворительной шейки матки, запоздалой беременности, холелитиаза, перерастяжения матки, стресса. Как описано более подробно далее, классификатор, описанный в настоящем описании, чувствителен к компоненту медицински показанных PTB на основании состояний, например, таких как предэклампсия или диабет беременных.
[00208] В некоторых вариантах осуществления настоящее раскрытие предусматривает биологические маркеры, пары биологических маркеров и/или обращения, приведенные здесь в качестве примера, с использованием ITIH4/CSH, которые являются мощными предикторами времени до родов (TTB) (фиг. 10). TTB определяют как разность между GABD и гестационным возрастом при рождении (GAB). Это исследование делает возможным предсказание TTB или GAB, индивидуально или при математической комбинации таких анализируемых веществ. Анализируемые вещества, которые не имеют различия между случаем и контролем, но демонстрируют изменения в интенсивности анализируемого вещества на протяжении беременности, можно использовать в хронологии беременности в соответствии со способами по изобретению. Калибровка множества анализируемых веществов, которые нельзя использовать для диагностирования преждевременных родов других нарушений, можно использовать для того, чтобы датировать беременность. Такая хронология беременности важна для того, чтобы подтверждать датирование с помощью другой меры (например, даты последнего менструального цикла и/или ультразвукового датирования), или ее можно использовать отдельно для того, чтобы впоследствии и более точно предсказывать, например, sPTB, GAB или TTB. Эти анализируемые вещества, также упоминаемые в настоящем описании как «хронологические белки», можно использовать для того, чтобы датировать беременность в отсутствие других способов датирования или в сочетании с ними. В таблице 60 предоставлен список хронологических белков, которые можно использовать в хронологии беременности по изобретению для того, чтобы предсказать TTB и GAB.
[00209] В дополнительных вариантах осуществления способы определения вероятности преждевременных родов у беременной женщины дополнительно включают обнаружение поддающейся измерению особенности для одного или нескольких признаков риска, связанных с преждевременными родами. В дополнительных вариантах осуществления признаки риска выбирают из группы, состоящей из предыдущей низкой массы при рождении или преждевременного родоразрешения, множественных самопроизвольных абортов 2-го триместра, предшествующего вызванного аборта первого триместра, семейных факторов и факторов различных поколений, анамнеза бесплодия, неспособности к деторождению, числа беременностей, первой беременности, повторной беременности, аномалий плаценты, аномалий матки и шейки матки, кровотечения при беременности, задержки внутриутробного развития, экспозиции диэтилстильбэстрола in utero, множественных беременностей, пола младенца, небольшого роста, низкой массы перед беременностью, низкого или высокого индекса массы тела, диабета, гипертензии и урогенитальных инфекций.
[00210] «Поддающаяся измерению особенность» представляет собой какое-либо свойство, характеристику или аспект, которые можно определять и коррелирвоать с вероятностью преждевременных родов у субъекта. Термин дополнительно включает какое-либо свойство, характеристику или аспект, которые можно определять и коррелировать в связи с предсказанием GAB, предсказанием полносрочных родов или предсказанием времени до родов у беременной женщины. Для биологического маркера такая поддающаяся измерению особенность может включать, например, присутствие, отсутствие или концентрацию биологического маркера или его фрагмента в биологическом образце, измененную структуру, например, такую как присутствие или количество посттрансляционных модификаций, таких как окисление, в одном или нескольких положениях в аминокислотной последовательности биологического маркера или, например, присутствие измененной конформации в сравнении с конформацией биологического маркера у полносрочного контрольного субъекта и/или присутствие, количество или измененная структура биологического маркера в качестве части профиля из больше чем одного биологического маркера.
[00211] В дополнение к биологическим маркерам, поддающиеся измерению особенности дополнительно могут включать признаки риска, включая, например, материнские характеристики, возраст, расу, этническую принадлежность, медицинский анамнез, анамнез прошлой беременности, акушерский анамнез. Для признака риска поддающаяся измерению особенность может включать, например, предыдущую низкую массу при рождении или преждевременное родоразрешение, множественные самопроизвольные аборты 2-го триместра, предшествующий вызванный аборт первого триместра, семейные факторы и факторы различных поколений, анамнез бесплодия, неспособность к деторождению, аномалии плаценты, аномалии матки и шейки матки, измерения малой длины шейки матки, кровотечение при беременности, задержку внутриутробного развития, экспозицию диэтилстильбэстрола in utero, множественные беременности, пол младенца, небольшой рост, низкую массу перед беременностью/низкий индекс массы тела, диабет, гипертензию, урогенитальные инфекции, гипотиреоидизм, астму, низкую образовательную подготовку, курение сигарет, использование лекарственных средств и употребление алкоголя.
[00212] В некоторых вариантах осуществления способы по изобретению включают вычисление индекса массы тела (BMI).
[00213] В некоторых вариантах осуществления раскрытые способы определения вероятности преждевременных родов включают обнаружение и/или количественное определение одного или нескольких биологических маркеров с использованием масс-спектрометрии, захватывающего средства или их сочетания.
[00214] В дополнительных вариантах осуществления раскрытые способы определения вероятности преждевременных родов у беременной женщины включают начальную стадию предоставления биологического образца от беременной женщины.
[00215] В некоторых вариантах осуществления раскрытые способы определения вероятности преждевременных родов у беременной женщины включают передачу вероятности поставщику медицинских услуг. Раскрытые предсказания GAB, способы предсказания полносрочных родов, способы определения вероятности полносрочных родов у беременной женщины, а также способы предсказания времени до родов у беременной женщины, аналогичным образом, включают передачу вероятности поставщику медицинских услуг. Как изложено выше, несмотря на то, что описаны и приведены в качестве примера со ссылкой на определение вероятности преждевременных родов у беременной женщины, все варианты осуществления, описанные на всем протяжении этого раскрытия, аналогичным образом, применимы к способам предсказания GAB, способам предсказания полносрочных родов, способам определения вероятности полносрочных родов у беременной женщины, а также способам предсказания времени до родов у беременной женщины. В частности, биологические маркеры и панели, перечисленные на всем протяжении этой заявки с прямым указанием на способы для преждевременных родов, также можно использовать в способах предсказания GAB, способах предсказания полносрочных родов, способах определения вероятности полносрочных родов у беременной женщины, а также способах предсказания времени до родов у беременной женщины. Специалисту в данной области будет ясно, что каждый из указанных выше способов имеет конкретные и существенные полезности и эффекты в отношении соображений здоровья матери и плода.
[00216] В дополнительных вариантах осуществления посредством связи информируют о последующем решении о лечении для беременной женщины. В некоторых вариантах осуществления способ определения вероятности преждевременных родов у беременной женщины включает дополнительную особенность - выражение вероятности в качестве оценки риска.
[00217] В способах, описанных в настоящем описании, определение вероятности преждевременных родов у беременной женщины включает начальную стадию, которая включает формирование показателя вероятности/риска посредством измерения соотношения выделенных биологических маркеров, выбранных из группы в когорте преждевременных беременностей и полносрочных беременностей с известным гестационным возрастом при родах. Для индивидуальной беременности определение вероятности преждевременных родов у беременной женщины включает измерение соотношения выделенного биологического маркера с использованием того же способа измерения, как используют на начальной стадии создания показателя вероятности/риска и сравнения измеряемого соотношения с показателем риска для того, чтобы получать персонализированный риск для индивидуальной беременности. В одном из вариантов осуществления показатель преждевременного риска формируют посредством измерения соотношения IBP4/SHBG в когорте преждевременных и полносрочных беременностей, где зарегистрирован гестационный возраст при рождении. Затем в клинической практике измеряемое соотношение IBP4/SHBG для индивидуальной беременности сравнивают с показателем для того, чтобы получать преждевременный риск, используя те же технологии выделения и измерения для получения IBP4/SHBG, как в основной группе.
[00218] Как используют в настоящем описании, термин «оценка риска» относится к оценке, присвоенной на основании сравнения количества одного или нескольких биологических маркеров или значений обращения в биологическом образце, полученном у беременной женщины, со стандартной или эталонной оценкой, которая представляет усредненное количество одного или нескольких биологических маркеров, вычисленное для биологических образцов, полученных из случайной совокупности беременных женщин. В некоторых вариантах осуществления оценку риска выражают в виде логарифма значения обращения, т. е. соотношения относительных интенсивностей индивидуальных биологических маркеров. Специалист в данной области примет во внимание, что оценку риска можно выражать на основании различных преобразований данных, а также можно выражать как само соотношение. Кроме того, конкретно в отношении пар обращения, специалист в данной области примет во внимание, что любое соотношение в равной мере информативно, если биологические маркеры в числителе и знаменателе поменяны, или что применены соответствующие преобразования данных (например, вычитание). Поскольку уровень биологического маркера может не быть статическим на всем протяжении беременности, стандартную или эталонную оценку следует получать для момента беременности, который соответствует таковому у беременной женщины во время взятия образца. Стандартную или эталонную оценку можно определять предварительно и встраивать в модель предиктора так, что сравнение является непрямым вместо фактического выполнения каждый раз, когда определяют вероятность для субъекта. Оценка риска может представлять собой стандарт (например, число) или порог (например, линию на графике). Значение оценки риска коррелирует с отклонением, вверх или вниз, от усредненного количества одного или нескольких биологических маркеров, вычисленного для биологических образцов, полученных из случайной совокупности беременных женщин. В определенных вариантах осуществления, если оценка риска больше стандартной или эталонной оценки риска, беременная женщина может иметь повышенную вероятность преждевременных родов. В некоторых вариантах осуществления величина оценки риска беременной женщины или количество, на которое она превышает эталонную оценку риска, может отражать уровень риска беременной женщины или коррелировать с ним.
[00219] Как показано на примере в настоящем описании, классификатор PreTRMTM определяют как натуральный логарифм SIS нормализованных интенсивностей перехода пептида IBP4 (QCHPALDGQR_394.5_475.2 (SEQ ID № 2)) и перехода пептида SHBG (IALGGLLFPASNLR_481.3_657.4 (SEQ ID № 18)). Оценка=ln(P1n/P2n), где P1n и P2n представляют собой значения SIS нормализованной площади пика для переходов IBP4 и SHBG, соответственно. SIS нормализацию определяют как удельное соотношение эндогенной площади пика, деленной на соответствующую SIS площадь пика: например P1n=P1e/P1SIS, где P1e=площадь пика для эндогенного перехода IBP4 и P1SIS=площадь пика для SIS перехода IBP4. По идентифицированной связи между распределением оценок PreTRMTM и соответствующему скорректированному по болезненности положительному прогностическому значению неизвестному субъекту можно присваивать вероятность sPTB на основании определения его оценки». Эта зависимость или связь представлена на фиг. 25, и она соединяет лабораториое измерение с клиническим прогнозом.
[00220] Хотя классификатор PreTRMTM определяют как натуральный логарифм SIS нормализованных интенсивностей перехода пептида IBP4 (QCHPALDGQR_394.5_475.2 (SEQ ID № 2)) и перехода пептида SHBG (IALGGLLFPASNLR_481.3_657.4 (SEQ ID № 18)), изобретение также содержит классификаторы, которые включают множество обращений. Повышенной эффективности можно достичь посредством конструирования предикторов, сформированных из больше чем одного обращения. Следовательно, в дополнительных вариантах осуществления изобретения способы включают множество обращений, которые обладают высокой предсказательной эффективностью, например, для отдельных интервалов GABD, преждевременного разрыва околоплодных оболочек (PPROM) в сравнении с преждевременными сокращениями матки в отсутствие PPROM (PTL), пола плода, первой беременности в сравнении с повторной беременностью. Этот вариант осуществления приведен в качестве примера в примере 10 и таблице 61 для любых обращений, которые давали высокую предсказательную эффективность в более раннем (например, недели 17-19) или более позднем (например, недели 19-21) диапазоне гестационного возраста. Как показано на примере, эффективность предикторов, формируемых из комбинаций (SumLog) множества обращений, оценивали для всего диапазона взятия крови, и оценку предиктора получали из суммирования значений Log индивидуального обращения (SumLog). Специалист в данной области может выбирать другие модели (например, логистическую регрессию) для того, чтобы строить предиктор, сформированный из больше чем одного обращения.
[00221] Способы по изобретению дополнительно включают классификаторы, которые содержат индикаторную переменную, которая выбирает один или поднабор обращений на основании известных клинических факторов, например, периода взятия крови, пола плода, числа беременностей, а также каких-либо других узнаваемых особенностей и/или факторов риска пациента, описанных на всем протяжении этой заявки. Этот вариант осуществления приведен в качестве примера в примере 10, таблицах с 61 до 64, которые иллюстрируют эффективность обращения (недели 17-21) независимо ддля двух различных фенотипов sPTB, PPROM и PTL. Этот вариант осуществления аналогичным образом иллюстрирован в примере 10, таблицах 76 и 77 и на фиг. 108 и 109, которые иллюстрируют эффективность обращения (недели 19-21) независимо для двух различных фенотипов sPTB, преждевременного разрыва околоплодных оболочек (PPROM) и преждевременных сокращений матки в отсутствие PPROM (PTL). Способы по изобретению, таким образом, включают отбор обращений для построениях независимых предикторов для PPROM и PTL или для максимизации общей эффективности с использованием комбинации из больше чем одного обращения в одном предикторе, как описано выше. Этот вариант осуществления дополнительно иллюстрирован в примере 10, таблицах 65-68, которые иллюстрируют эффективность обращения (недели 17-21) независимо для двух различных типов sPTB, первой беременности и повторной беременности. Этот вариант осуществления дополнительно иллюстрирован в примере 10, таблицах 69-72 и на фиг. 106, которые иллюстрируют эффективность обращения (недели 17-21) независимо для двух различных типов sPTB на основании пола плода. Хотя приведены примеры в отношении PPROM и PTL, числа беременностей и пола плода, способы по изобретению включают классификаторы, которые содеражт индикаторную переменную, которая выбирает одно или поднабор обращений на основании GABD или каких-либо известных клинических факторов/факторов риска, описанных в настоящем описании или иным образом известных специалистам в данной области. В качестве альтернативы классификатору, который содержит индикаторную переменную, изобретение дополнительно предусматривает раздельные классификаторы, которые подгоняют к поднаборам беременных женщин на основании GABD или каких-либо известных клинических факторов/факторов риска, описанных в настоящем описании или иным образом известных специалистам в данной области. Например, этот вариант осуществления охватывает раздельные классификаторы для последовательных и/или перекреывающихся временных интервалов для GABD, которые основаны на наиболее эффективных обращениях для каждого временного интервала.
[00222] Как показано на примере в настоящем описании, предсказательную эффективность заявленных способов можно усовершенствовать с использованием BMI-стратификации > 22 и≤37 кг/м2. Соответственно, в некоторых вариантах осуществления способы по изобретению можно осуществлять на практике с использованием образцов, полученных от беременных женщин с конкретным BMI. В кратком изложении BMI представляет собой массу индивидуума в килограммах, деленную на квадрат роста в метрах. BMI не измеряет телесный жир непосредственно, но исследования показали, что BMI коррелирует с более прямыми мерами для телесного жира, получаемыми из измерений толщины кожной складки, биоэлектрического импеданса, денситометрии (подводного взвешивания), двухэнергетической рентгеновской абсорбциометрии (DXA) и других способов. Кроме того, BMI, по-видимому, также сильно коррелирует с различными метаболическими и патологическими исходами, как эти более прямые меры для телесного жира. В целом, индивидуума с BMI ниже 18,5 считают имеющим недостаточный вес, индивидуума с BMI от≥18,5 до 24,9 имеющим нормальный вес, тогда как индивидуума с BMI от≥25,0 до 29,9 считают имеющим избыточный вес и индивидуума с BMI≥30,0 считают имеющим ожирение. В некоторых вариантах осуществления предсказательную эффективность заявленных способов можно усовершенствовать с использованием BMI-стратификации≥18, ≥19, ≥20, ≥21, ≥22, ≥23, ≥24, ≥25, ≥26, ≥27, ≥28, ≥29 или≥30. В других вариантах осуществления предсказательную эффективность заявленных способов можно усовершенствовать с использованием BMI-стратификации≤18, ≤19, ≤20, ≤21, ≤22, ≤23, ≤24, ≤25, ≤26, ≤27, ≤28, ≤29 или≤30.
[00223] В контексте настоящего изобретения термин «биологический образец» охватывает любой образец, который получен у беременной женщины и содержит один или несколько биологических маркеров, описанных в настоящем описании. Подходящие образцы в контексте настоящего изобретения включают, например, кровь, плазму, сыворотку, амниотическую жидкость, вагинальные выделения, слюну и мочу. В некоторых вариантах осуществления биологический образец выбирают из группы, состоящей из цельной крови, плазмы и сыворотки. В конкретном варианте осуществления биологический образец представляет собой сыворотку. Специалисты в данной области примут во внимание, что биологический образец может содержать какую-либо фракцию или компонент крови, без ограничения T-клетки, моноциты, нейтрофилы, эритроциты, тромбоциты и микровезикулы, такие как экзосомы и экзосомоподобные везикулы. В конкретном варианте осуществления биологический образец представляет собой сыворотку.
[00224] Как используют в настоящем описании, термин «преждевременные роды» относится к родоразрешению или родам при гестационном возрасте меньше чем 37 полных недел. Установлены другие широко используемые подкатегории преждевременных родов, которые описывают умеренно преждевременные роды (с 33 до 36 недель беременности), очень преждевременные роды (<33 недель беременности) и чрезвычайно преждевременные роды (≤28 недель беременности). По отношению к способам, описанным в настоящем описании, специалистам в данной области будет понятно, что пороги, которые описывают преждевременные роды и полносрочные роды, а также пороги, которые описывают подкатегории преждевременных родов, можно корректировать при практическом осуществлении способов, описанных в настоящем описании, например, для того, чтобы максимизировать конкретный эффект для здоровья. В различных вариантах осуществления изобретения, порог, который описывает преждевременные роды, включаюет, например, рождение в≤37 недель беременности, ≤36 недель беременности, ≤35 недель беременности, ≤34 недель беременности, ≤33 недель беременности, ≤32 недель беременности, ≤30 недель беременности, ≤29 недель беременности, ≤28 неделт беременности, ≤27 недель беременности, ≤26 недель беременности, ≤25 недель беременности, ≤24 недель беременности, ≤23 недель беременности или≤22 недель беременности. В некоторых вариантах осуществления порог, описывающий преждевременные роды, составляет≤35 недель беременности. Кроме того, понятно, что такие корректировки составляют навыки индивидуумов, которых считают специалистами в данной области, и они включены в объем изобретения, описанный в настоящем описании. Гестационный возраст является заменой для степени развития плода и готовности плода к родам. Гестационный возраст обычно определяют как длительность времени от даты последних нормальных месячных до даты родов. Однако, акушерские меры и ультразвуковые оценки также могут содействовать оцениванию гестационного возраста. Преждевременные роды в целом разделяют на две отдельные подгруппы. Первая - самопроизвольные преждевременные роды, представляющие собой то, что происходит после самопроизвольного начала преждевременных сокращений матки или преждевременного разрыва околоплодных оболочек независимо от последующей стимуляции родов или родоразрешения кесаревым сечением. Вторая - медицински показанные преждевременные роды, представляющие собой то, что происходит после индукции или кесарева сечения, для одного или нескольких состояний, которое лицо, осуществляющее уход за женщиной, определяет как угрожающее здоровью или жизни матери и/или плода. В некоторых вариантах осуществления способы, описанные в настоящем описании, направлены на определение вероятности самопроизвольных преждевременных родов или медицински показанных преждевременных родов. В некоторых вариантах осуществления способы, описанные в настоящем описании, направлены на определение вероятности для самопроизвольных преждевременных родов. В дополнительных вариантах осуществления способы, описанные в настоящем описании, направлены на медицински показанные преждевременные роды. В дополнительных вариантах осуществления способы, описанные в настоящем описании, направлены на предсказание гестационного возраста при рождении.
[00225] Как используют в настоящем описании, термин «оценочный гестационный возраст» или «оценочный GA» относится к GA, определяемому на основании даты последних нормальных месячных и дополнительных акушерских измерений, ультразвуковых оценок или других клинических параметров, включая, без ограничения, те, которые описаны в предшествующем параграфе. В отличие от этого, термин «предсказанный гестационный возраст при рождении» или «предсказанный GAB» относится к GAB, определяемому на основании способов по изобретению, как раскрыто в настоящем описании. Как используют в настоящем описании, «полносрочные роды» относится к родам при гестационном возрасте≥37 полных недель.
[00226] В некоторых вариантах осуществления беременная женщина находится между 17 и 28 неделями беременности в момент взятия биологического образца, также обозначаемый как GABD (гестационный возраст при взятии крови). В других вариантах осуществления беременная женщина находится между 16 и 29 неделями, между 17 и 28 неделями, между 18 и 27 неделями, между 19 и 26 неделями, между 20 и 25 неделями, между 21 и 24 неделями или между 22 и 23 неделями беременности в момент взятия биологического образца. В дополнительных вариантах осуществления беременная женщина находится между приблизительно 17 и 22 неделями, между приблизительно 16 и 22 неделями, между приблизительно 22 и 25 неделями, между приблизительно 13 и 25 неделями, между приблизительно 26 и 28 или между приблизительно 26 и 29 неделями беременности в момент взятия биологического образца. Соответственно, гестационный возраст беременной женщины в момент взятия биологического образца может составлять 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 недель. В конкретных вариантах осуществления биологический образец берут между 19 и 21 неделями гестационного возраста. В конкретных вариантах осуществления биологический образец берут между 19 и 22 неделями гестационного возраста. В конкретных вариантах осуществления биологический образец берут между 19 и 21 неделями гестационного возраста. В конкретных вариантах осуществления биологический образец берут между 19 и 22 неделями гестационного возраста. В конкретных вариантах осуществления биологический образец берут на 18 неделе гестационного возраста. В дополнительных вариантах осуществления наиболее эффективные обращения для последовательных или перекрывающихся временных интервалов можно комбинировать в одном классификаторе для того, чтобы предсказывать вероятность sPTB в более широком интервале гестационного возраста при взятии крови.
[00227] Термин «количество» или «уровень», как используют в настоящем описании, относится к количеству биологического маркера, который поддается обнаружению или поддается измерению в биологическом образце и/или контроле. Количество биологического маркера может представлять собой, например, количество полипептида, количество нуклеиновой кислоты или количество фрагмента или суррогата. Альтернативно термин может включать их сочетания. Термин «количество» или «уровень» биологического маркера представляет собой поддающуюся измерению особенность этого биологического маркера.
[00228] Изобретение также относится к способу обнаружения пары выделенных биологических маркеров, выбранных из группы, состоящей из пар биологических маркеров, точно определенных в какой-либо из таблиц с 1 до 77 и на фиг. с 1 до 111, у беременной женщины, указанный способ включает стадии a. получения биологического образца от беременной женщины; b. обнаружения присутствия пары выделенных биологических маркеров в биологическом образце посредством контакта биологического образца с первым захватывающим средством, которое специфически связывает первый элемент указанной пары, и вторым захватывающим средством, которое специфически связывает второй элемент указанной пары; и обнаружения связывания между первым биологическим маркером указанной пары и первым захватывающим средством и между вторым элементом указанной пары и вторым захватывающим средством.
[00229] Изобретение также относится к способу обнаружения пары выделенных биологических маркеров, выбранных из группы, состоящей из пар биологических маркеров, которые точно определены в таблицах с 27 до 59, с 61 до 72, 76 и 77, у беременной женщины, указанный способ включает стадии a. получения биологического образца от беременной женщины; b. обнаружения присутствия пары выделенных биологических маркеров в биологическом образце посредством контакта биологического образца с первым захватывающим средством, которое специфически связывает первый элемент указанной пары, и вторым захватывающим средством, которое специфически связывает второй элемент указанной пары; и обнаружения связывания между первым биологическим маркером указанной пары и первым захватывающим средством и между вторым элементом указанной пары и вторым захватывающим средством.
[00230] Изобретение также относится к способу обнаружения пары выделенных биологических маркеров, выбранной из группы, состоящей из IBP4/SHBG, VTNC/VTDB, VTNC/SHBG, CATD/SHBG, PSG2/ITIH4, CHL1/ITIH4, PSG2/C1QB, PSG2/FBLN3, HPX/IBP4 и HPX/PTGDS, у беременной женщины, указанный способ включает стадии a. получения биологического образца от беременной женщины; b. обнаружения присутствия пары выделенных биологических маркеров в биологическом образце посредством контакта биологического образца с первым захватывающим средством, которое специфически связывает первый элемент указанной пары, и вторым захватывающим средством, которое специфически связывает второй элемент указанной пары; и обнаружения связывания между первым биологическим маркером указанной пары и первым захватывающим средством и между вторым элементом указанной пары и вторым захватывающим средством. В одном из вариантов осуществления изобретение предусматривает способ обнаружения IBP4 и SHBG у беременной женщины, указанный способ включает стадии
a. получения биологического образца от беременной женщины; b. обнаружения присутствия IBP4 и SHBG в биологическом образце посредством контакта биологического образца с захватывающим средством, которое специфически связывает IBP4, и захватывающим средством, которое специфически связывает SHBG; и c. обнаружения связывания между IBP4 и захватывающим средством и между SHBG и захватывающим средством. В одном из вариантов осуществления способ включает измерение значения обращения для пары биологических маркеров. В дополнительном варианте осуществления существование изменения значения обращения между беременной женщиной и полносрочным контролем отражает вероятность преждевременных родов у беременной женщины. В одном из вариантов осуществления образец получают между 19 и 21 неделями гестационного возраста. В дополнительном варианте осуществления захватывающее средство выбирают из группы, состоящей из и антитела, фрагмента антитела, реактива на основе нуклеиновой кислоты, связывающего белка, низкомолекулярного соединения или его варианта. В дополнительном варианте осуществления способ осуществляют с помощью анализа, выбранного из группы, состоящей из иммуноферментного анализа (EIA), твердофазного иммуноферментного анализа (ELISA) и радиоиммунного анализа (RIA).
[00231] Изобретение также относится к способу обнаружения пары выделенных биологических маркеров, выбранной из группы, состоящей из IBP4/SHBG, VTNC/VTDB, VTNC/SHBG, CATD/SHBG, PSG2/ITIH4, CHL1/ITIH4, PSG2/C1QB, PSG2/FBLN3, HPX/IBP4 и HPX/PTGDS, у беременной женщины, указанный способ включает стадии a. получения биологического образца от беременной женщины; и b. обнаружения присутствия пары выделенных биологических маркеров в биологическом образце, которое включает применение образца на протеомном технологическом маршруте, состоящем из масс-спектрометрического количественного определения.
[00232] В одном из вариантов осуществления изобретение относится к способу обнаружения IBP4 и SHBG у беременной женщины, указанный способ включает стадии a. получения биологического образца от беременной женщины; и b. обнаружения присутствия пары выделенных биологических маркеров в биологическом образце, которое включает применение образца на протеомном технологическом маршруте, состоящем из масс-спектрометрического количественного определения.
[00233] «Протеомный технологический маршрут» в целом охватывает одну или несколько из следующих стадий: образцы сыворотки оттаивают и истощают 14 белков с наибольшим относительным содержанием посредством иммунной аффинной хроматографии. Истощенную сыворотку расщепляют протеазой, например, трипсином, чтобы получать пептиды. Затем расщепленную сыворотку обогащают смесью SIS пептидов и затем обессоливают и проводят LC-MS/MS с тройным квадрупольным прибором, работающим в режиме MRM. Коэффициенты ответа формируют из соотношений площадей пиков эндогенных пептидов и соответствующих эквивалентных пиков SIS пептидов. Специалисты в данной области примут во внимание, что в способах по изобретению можно использовать MS других типов, например, такую как MALDI-TOF или ESI-TOF. Кроме того, специалист в данной области может модифицировать протеомный технологический маршрут, например, выбирая конкретные реактивы (такие как протеазы) или опуская или изменяя порядок определенных стадий, например, может не быть необходимым иммунное истощение, SIS пептид можно добавлять раньше или позже, а меченные стабильными изотопами белки можно использовать в качестве стандартов вместо пептидов.
[00234] Любые существующие, доступные или стандартные способы разделения, обнаружения и количественного определения можно использовать в настоящем описании для того, чтобы измерять присутствие или отсутствие (например, считывание присутствия в сравнении с отсутствием; или поддающееся обнаружению количество в сравнении с не поддающимся обнаружению количеством) и/или количество (например, считывание абсолютного или относительного количества, например, такое как абсолютная или относительная концентрация) биологических маркеров, пептидов, полипептидов, белков и/или их фрагментов и необязательно одного или нескольких других биологических маркеров или их фрагментов в образцах. В некоторых вариантах осуществления обнаружение и/или количественное определение одного или нескольких биологических маркеров включает анализ, в котором используют захватывающее средство. В дополнительных вариантах осуществления захватывающее средство представляет собой антитело, фрагмент антитела, реактив на основе нуклеиновой кислоты, связывающий белок, низкомолекулярное соединение или его вариант. В дополнительных вариантах осуществления анализ представляет собой иммуноферментный анализ (EIA), твердофазный иммуносорбентный анализ (ELISA) и радиоиммунный анализ (RIA). В некоторых вариантах осуществления обнаружение и/или количественное определение одного или нескольких биологических маркеров дополнительно включает масс-спектрометрию (MS). В других вариантах осуществления масс-спектрометрия представляет собой коиммунопреципитацию-масс-спектрометрияю (co-IP MS), где за коиммунопреципитацией, способом, подходящим для выделения целых белковых комплексов, следует масс-спектрометрический анализ.
[00235] Как используют в настоящем описании, термин «масс-спектрометр» относится к устройству, способному улетучивать/ионизировать анализируемые вещества для того, чтобы формировать ионы газовой фазы и определять их абсолютные или относительные молекулярные массы. Подходящие способы улетучивания/ионизации представляют собой ионизацию лазерной десорбцией с использованием матрицы (MALDI), электрораспыление, лазер/свет, тепловое, электрическое, атомизированное/распыленное и т. п., или их сочетания. Подходящме формы масс-спектрометрии включают, но не ограничиваясь этим, приборы с ионной ловушкой, квадрупольные приборы, электростатические и магнитные секторные приборы, времяпролетные приборы, времяпролетный тандемный масс-спектрометр (TOF MS/MS), масс-спектрометры с преобразованием Фурье, Orbitrap и гибридные приборы, состоящие из различных комбинаций масс-анализаторов этих типов. Эти приборы, в свою очередь, можно сопрягать с различными другими приборами, которые фракционируют образцы (например, способы жидкостной хроматографии или твердофазной адсорбции на основании химических или биологических свойств) и которые ионизируют образцы для введения в масс-спектрометр, включая ионизацию лазерной десорбцией с использованием матрицы (MALDI), электрораспыление или ионизацию нанораспылением (ESI) или их сочетания.
[00236] В целом, любой масс-спектрометрический (MS) способ, который может предоставлять точную информацию о массе пептидов и также предпочтительно о фрагментировании и/или (частичной) аминокислотной последовательности выбранных пептидов (например, тандемная масс-спектрометрия, MS/MS; или с распадом за пределами источника, TOF MS), можно использовать в способах, описанных в настоящем описании. Подходящие пептидные способы MS и MS/MS и системы общеизвестны per se (см., например, Methods in Molecular Biology, том 146: «Mass Spectrometry of Proteins and Peptides», ред. Chapman, Humana Press 2000; Biemann 1990. Methods Enzymol 193: 455-79; или Methods in Enzymology, том 402: «Biological Mass Spectrometry», Burlingame, ред., Academic Press 2005), и их можно использовать при практическом осуществлении способов, описанных в настоящем описании. Соответственно, в некоторых вариантах осуществления раскрытые способы включают выполнение количественной MS для того, чтобы измерять один или несколько биологических маркеров. Такие количественные способы можно осуществлять в автоматизированном (Villanueva, et al., Nature Protocols (2006) 1(2):880-891) или полуавтоматизированном формате. В конкретных вариантах осуществления MS может быть функционально связан с устройством жидкостной хроматографии (LC-MS/MS или LC-MS) или устройством газовой хроматографии (GC-MS или GC-MS/MS). Другие способы, которые можно использовать в этом контексте, включают аффинные метки с изотопным кодированием (ICAT), тандемные массовые метки (TMT) или мечение стабильными изотопами с использованием аминокислот в клеточной культуре (SILAC), после чего следует хроматография и MS/MS.
[00237] Как используют в настоящем описании, термины «мониторинг нескольких реакций (MRM)» или «мониторинг выбранных реакций (SRM)» относятся к основанному на MS способу количественного определения, который, в частности, можно использовать для количественного определения анализируемых веществ, которые имеют низкое относительное содержание. В SRM эксперименте предварительно определяемый ион-предшественник и один или несколько его фрагментов выбирают с помощью двух фильтров массы тройного квадрупольного прибора и осуществляют мониторинг с течением времени для точного количественного определения. Несколько пар SRM предшественников и фрагментарных ионов можно измерять в одном и том же эксперименте в масштабе хроматографического времени посредством быстрого переключения между различными парами предшественников/фрагментов для того, чтобы осуществлять MRM эксперимент. Серия переходов (пар предшественника/фрагментарного иона) в комбинации со временем удержания целевого анализируемого вещества (например, пептида или низкомолекулярного соединения, такого как химическая частица, стероид, гормон) может составлять окончательный анализ. Можно определять количество большого числа анализируемых веществ во время одного LC-MS эксперимента. Термин «запланированный» или «динамический», в отношении MRM или SRM, связан с вариацией анализа, в котором переходы для конкретного анализируемого вещества регистрируют только во временном интервале около ожидаемого времени удержания, значительно увеличивая число анализируемых веществ, которые можно обнаруживать и определять количественно в одном LC-MS эксперименте и содействуя избирательности теста, поскольку время удержания представляет собой свойство, зависящее от физической природы анализируемого вещества. Также можно осуществлять мониторинг одного анализируемого вещества с использованием больше чем одного перехода. Наконец, в анализ можно включать стандарты, которые соответствуют анализируемым веществам, представляющим интерес (например, ту же аминокислотную последовательность), которые отличаются включением стабильных изотопов. Стабильные изотопные стандарты (SIS) можно внедрядть в анализ в точных количествах и использовать для того, чтобы количественно определять соответствующее известное анализируемое вещество. Дополнительный уровень специфичности обеспечивают посредством совместного элюирования неизвестного анализируемого вещества и соответствующешго ему SIS и свойств их переходов (например, сходство в соотношении уровня двух переходов неизвестного и соотношении двух переходов соответствующего ему SIS).
[00238] Масс-спектрометрические анализы, приборы и системы, подходящие для анализа пептидов биологических маркеров, могут включать, без ограничения, времяпролетную MS при ионизации лазерной десорбцией с использованием матрицы (MALDI-TOF); MALDI-TOF с распадом за пределами источника (PSD); MALDI-TOF/TOF; времяпролетную масс-спектрометрию усиленной поверхностью лазерной десорбции/ионизации (SELDI-TOF MS); масс-спектрометрию с ионизацией электрораспылением (ESI-MS); ESI-MS/MS; ESI-MS/(MS)n (n представляет собой целое число больше нуля); ESI 3D или линейную (2D) MS с ионной ловушкой; ESI тройную квадрупольную MS; ESI квадрупольное ортогональное TOF (Q-TOF); ESI MS системы с преобразованием Фурье; десорбцию/ионизацию на кремнии (DIOS); масс-спектрометрию вторичных ионов (SIMS); масс-спектрометрию с химической ионизацией при атмосферном давлении (APCI-MS); APCI-MS/MS; APCI-(MS)n; спектрометрию подвижности ионов (IMS); индукционно-связанно-плазменную масс-спектрометрию (ICP-MS); масс-спектрометрию с фотоионизацией при атмосферном давлении (APPI-MS); APPI-MS/MS; и APPI-(MS)n. Фрагментирования пептидных ионов в компоновках для тандемной MS (MS/MS) можно достигать, используя способы, устоявшиеся в данной области, такие как, например, индуцированная столкновениями диссоциация (CID). Как раскрыто в настоящем описании, обнаружение и количественное определение биологических маркеров посредством масс-спектрометрии может включать мониторинг нескольких реакций (MRM), такой как описано, среди прочих, у Kuhn et al. Proteomics 4: 1175-86 (2004). Регистрация в режиме запланированного мониторинга нескольких реакций (запланированный MRM) во время LC-MS/MS анализа повышает чувствительность и точность количественного определения пептидов. Anderson and Hunter, Molecular and Cellular Proteomics 5(4):573 (2006). Как раскрыто в настоящем описании, анализ на основе масс-спектрометрии можно благоприятно комбинировать со способами разделения или фракционирования пептидов или белков выше по технологическому потоку, например, такими как с использованием хроматографических и других способов, описанных в настоящем описании далее. Как дополнительно описано в настоящем описании, количественную протеомику способом дробовика можно комбинировать с анализом на основе SRM/MRM для высокопропускной идентификации и верификации прогностических биологических маркеров преждевременных родов.
[00239] Специалист в данной области примет во внимание, что множество способов можно использовать для того, чтобы определять количество биологического маркера, включая масс-спектрометрические подходы, такие как MS/MS, LC-MS/MS, мониторинг нескольких реакций (MRM) или SRM и мониторинг ионов-продуктов (PIM) и также включая способы на основе антител, такие как иммунологические анализы, такие как вестерн-блоттинг, твердофазный иммуносорбентный анализ (ELISA), иммунопреципитация, иммуногистохимия, иммунофлуоресценция, радиоиммунный анализ, дот-блоттинг и FACS. Соответственно, в некоторых вариантах осуществления определение уровня по меньшей мере одного биологического маркера включает использование иммунологического анализа и/или масс-спектрометрических способов. В дополнительных вариантах осуществления масс-спектрометрические способы выбирают из MS, MS/MS, LC-MS/MS, SRM, PIM и других таких способов, которые известны в данной области. В других вариантах осуществления LC-MS/MS дополнительно содержит 1D LC-MS/MS, 2D LC-MS/MS или 3D LC-MS/MS. Способы иммунологического анализа и протоколы в целом известны специалистам в данной области (Price and Newman, Principles and Practice of Immunoassay, 2-е изд., Grove's Dictionaries, 1997; и Gosling, Immunoassays: A Practical Approach, Oxford University Press, 2000). Можно использовать различные способы иммунологического анализа, включая конкурентные и не конкурентные иммунологические анализы (Self et al., Curr. Opin. Biotechnol., 7:60-65 (1996).
[00240] В дополнительных вариантах осуществления иммунологический анализ выбирают из вестерн-блоттинга, ELISA, иммунопреципитации, иммуногистохимии, иммунофлуоресценции, радиоиммунного анализа (RIA), дот-блоттинга и FACS. В определенных вариантах осуществления иммунологический анализ представляет собой ELISA. В еще одном дополнительном варианте осуществления ELISA представляет собой прямой ELISA (твердофазный иммуносорбентный анализ), непрямой ELISA, сэндвич ELISA, конкурентный ELISA, мультиплексный ELISA, технологии ELISPOT и другие схожие способы, известные в данной области. Принципы этих способов иммунологического анализа известны в данной области, например, John R. Crowther, The ELISA Guidebook, 1-е изд., Humana Press 2000, ISBN 0896037282. Обычно ELISA осуществляют с использованием антител, но их можно осуществлять с использованием каких-либо захватывающих средств, которые специфически связываются с одним или несколькими биологическими маркерами по изобретению и которые поддаются обнаружению. Мультиплексный ELISA делает возможным одновременное обнаружение двух или больше анализируемых веществ в одном компартменте (например, лунке микропланшета), обычно с множеством адресов массива (Nielsen and Geierstanger 2004. J Immunol Methods 290: 107-20 (2004) и Ling et al. 2007. Expert Rev Mol Diagn 7: 87-98 (2007)).
[00241] В некоторых вариантах осуществления радиоиммунный анализ (RIA) можно использовать для того, чтобы обнаруживать один или несколько биологических маркеров в способах по изобретению. RIA представляет собой конкурентный анализ, который хорошо известен в данной области и включает смешивание известных количеств радиоактивно-меченного (например, меченного 125I или 131I) целевого анализируемого вещества с антителом со специфичностью к анализируемому веществу, после чего следует добавление не меченного анализируемого вещества из образца и измерение количества меченного анализируемого вещества, которое вытеснено (см., например, An Introduction to Radioimmunoassay and Related Techniques, под ред. Chard T, Elsevier Science 1995, ISBN 0444821198 для руководства).
[00242] Поддающуюся обнаружению метку можно использовать в анализах, описанных в настоящем описании, для прямого или опосредованного обнаружения биологических маркеров в способах по изобретению. Широкий спектр поддающихся обнаружению меток можно использовать, выбирая метку в зависимости от необходимой чувствительности, простоты конъюгации с антителом, требований к стабильности и доступных приборов и мероприятий по утилизации. Специалисты в данной области знакомы с выбором подходящих поддающихся обнаружению меток на основании обнаружения биологических маркеров в анализах в способах по изобретению. Подходящие поддающиеся обнаружению метки включают, но не ограничиваясь этим, флуоресцентные красители (например, флуоресцеин, флуоресцеинизотиоцианат (FITC), Oregon Green™, родамин, техасский красный, тетрародимин изотиоцинат (TRITC), Cy3, Cy5 и т. д.), флуоресцентные маркеры (например, зеленый флуоресцентный белок (GFP), фикоэритрин и т. д.), ферменты (например, люциферазу, пероксидазу хрена, щелочную фосфатазу и т. д.), наночастицы, биотин, дигоксигенин, металлы и т. п.
[00243] Для анализа на основе масс-спектрометрии дифференциальное мечение с использованием изотопных реактивов, например, аффинных меток с изотопным кодированием (ICAT), или более новой вариации, в которой используют реактивы для изобарического мечения, iTRAQ (Applied Biosystems, Foster City, Calif.), или тандемных массовых меток, TMT, (Thermo Scientific, Rockford, IL), после чего следует многомерная жидкостная хроматография (LC) и анализ тандемной масс-спектрометрии (MS/MS) может предоставлять дополнительный способ при практическом осуществлении способов по изобретению.
[00244] Хемилюминесцентный анализ с использованием хемилюминесцентного антитела можно использовать для чувствительного, не радиоактивного обнаружения уровней белков. Антитело, меченное флуорохромром, также может подходить. Примеры флуорохромов включают, без ограничения, DAPI, флуоресцеин, Hoechst 33258, R-фикоцианин, B-фикоэритрин, R-фикоэритрин, родамин, техасский красный и лиссамин. Непрямые метки включают различные ферменты, хорошо известные в данной области, такие как пероксидаза хрена (HRP), щелочная фосфатаза (AP), β-галактозидаза, уреаза и т. п. Системы обнаружения с использованием подходящих субстратов для пероксидазы хрена, щелочной фосфатазы и β-галактозидазы хорошо известны в данной области.
[00245] Сигнал от прямой или непрямой метки можно анализировать, например, с использованием спектрофотометра для того, чтобы обнаруживать окраску хромогенного субстрата; счетчика излучения для того, чтобы обнаруживать излучение, например, γ-счетчик для обнаружения 125I; или флуорометр для того, чтобы обнаруживать флуоресценцию в присутствии света определенной длины волны. Для обнаружения антител, связанных с ферментами, можно выполнять количественный анализ с использованием спектрофотометра, такого как считыватель микропланшетов EMAX (Molecular Devices; Menlo Park, Calif.) в соответствии с инструкциями производителя. При желании, выполнение анализов, используемых для реализации изобретения на практике, можно автоматизировать или роботизировать, и сигнал от нескольких образцов можно обнаруживать одновременно.
[00246] В некоторых вариантах осуществления способы, описанные в настоящем описании, охватывают количественное определение биологических маркеров с использованием масс-спектрометрии (MS). В дополнительных вариантах осуществления масс-спектрометрия может представлять собой жидкостную хроматографию-масс-спектрометрию (LC-MS), мониторинг нескольких реакций (MRM) или мониторинг выбранных реакций (SRM). В дополнительных вариантах осуществления MRM или SRM дополнительно может охватывать запланированный MRM или запланированный SRM.
[00247] Как описано выше, хроматографию также можно использовать при практическом осуществлении способов по изобретению. Хроматография включает способы разделения химических веществ и в целом включает процесс, в котором движущийся поток жидкости или газа («подвижная фаза») несет смесь анализируемых веществ и разделеяет ее на компоненты в результате дифференциального распределения анализируемых веществ, когда они протекают через стационарную жидкую или твердую фазу («стационарную фазу») или около нее, между подвижной фазой и указанной стационарной фазой. Стационарная фаза обычно может представлять собой тонкодисперсное твердое вещество, лист фильтровального материала или тонкую пленку из жидкости на поверхности твердого вещества или тому подобное. Хроматография хорошо известна специалистам в данной области в качестве способа, который применяют для разделения химических соединений биологического происхождения, например, таких как аминокислоты, белки, фрагменты белков или пептидов и т. д.
[00248] Хроматография может быть колоночной (т. е., при которой стационарную фазу располагают или упаковывают в колонку), предпочтительно жидкостной хроматографией и еще более предпочтительно высокоэффективной жидкостной хроматографией (ВЭЖХ), или сверхвысокоэффективной жидкостной хроматографией/жидкостной хроматографией сверхвысокого давления (UHPLC). Подробности о хроматографии хорошо известны в данной области (Bidlingmeyer, Practical HPLC Methodology and Applications, John Wiley & Sons Inc., 1993). Образцовые типы хроматографии включают, без ограничения, высокоэффективную жидкостную хроматографию (ВЭЖХ), UHPLC, ВЭЖХ с нормальной фазой (NP-HPLC), ВЭЖХ с обращенной фазой (RP-HPLC), ионообменную хроматографию (IEC), например, катионо- или анионообменную хроматографию, хроматографию гидрофильного взаимодействия (HILIC), хроматографию гидрофобного взаимодействия (HIC), эксклюзионную хроматографию (SEC), включая гельфильтрационную хроматографию или гельпроникающую хроматографию, хроматофокусирование, аффинную хроматографию, такую как иммуноаффинность, аффинную хроматографию на иммобилизованном металле и т. п. Хроматографию, включая одномерну, двухмерную или большей размерности хроматографию, можно использовать в качестве способа фракционирования пептидов в сочетании с дополнительным способом анализа пептидов, например, таким как масс-спектрометрический анализ ниже по технологическому потоку, как описано в другом месте в данном описании.
[00249] Кроме того, можно использовать способы разделения, идентификации или количественного определения пептидов или полипептидов, необязательно в сочетании с каким-либо из описанных выше способов анализа, для измерения биологических маркеров в настоящем раскрытии. Такие способы включают, без ограничения, разделение химическим экстрагированием, изоэлектрическое фокусирование (IEF), включая капиллярное изоэлектрическое фокусирование (CIEF), капиллярный изотахофорез (CITP), капиллярную электрохроматографию (CEC) и т. п., одномерный электрофорез в полиакриламидном геле (PAGE), двухмерный электрофорез в полиакриламидном геле (2D-PAGE), капиллярный электрофорез в геле (CGE), капиллярный зонный электрофорез (CZE), мицеллярную электрокинетическую хроматографию (MEKC), электрофорез в свободном потоке (FFE) и т. д.
[00250] В контексте изобретения термин «захватывающее средство» относится к соединению, которое может специфически связываться с мишенью, в частности, с биологическим маркером. Термин включает антитела, фрагменты антител, реактивы на основе нуклеиновой кислоты, связывающей белок (например, аптамеры, Slow Off-rate Modified Aptamers (SOMAmer™)), средства, которые захватывают белки, природные лиганды (т. е. гормон для его рецептора или наоборот), низкомолекулярные соединения или их варианты.
[00251] Захватывающие средства можно конфигурировать для того, чтобы специфически связывать мишень, в частности, биологический маркер. Захватывающие средства могут включать, но не ограничиваясь этим, органические молекулы, такие как полипептиды, полинуклеотиды и другие неполимерные молекулы, которые может идентифицировать специалист. В вариантах осуществления, описанных в настоящем описании, захватывающие средства включают какое-либо средство, которое можно использовать для того, чтобы обнаруживать, очищать, выделять или обогащать мишень, в частности, биологический маркер. Можно использовать любые известные в данной области технологии аффинного захвата для того, чтобы избирательно выделять и обогащать/концентрировать биологические маркеры, которые представляют собой компоненты сложных смесей биологических сред, для использования в раскрытых способов.
[00252] Антительные захватывающие средства, которые специфически связываются с биологическим маркером, можно получать с использованием любых подходящих известных в данной области способов. См., например, Coligan, Current Protocols in Immunology (1991); Harlow & Lane, Antibodies: A Laboratory Manual (1988); Goding, Monoclonal Antibodies: Principles and Practice (2-е изд. 1986). Антительные захватывающие средства могут представлять собой любой иммуноглобулин или его производное, которые получены из природы или полностью или частично синтетически. Все их производные, которые сохраняют способность к специфическому связыванию, также включены в термин. Антительные захватывающие средства имеют связывающий домен, который гомологичен или главным образом гомологичен иммуноглобулиновому связывающему домену и может быть получен из природных источников или получен частично или полностью синтетически. Антительные захватывающие средства могут представлять собой моноклональные или поликлональные антитела. В некоторых вариантах осуществления антитело представляет собой одноцепочечное антитело. Средние специалисты в данной области примут во внимание, что антитела можно предоставлять в любой из различных форм, включая, например, гуманизированную, частично гуманизированную, химерную, химерную гуманизированную и т. д. Антительные захватывающие средства могут представлять собой фрагменты антител, включая в качестве неограничивающих примеров Fab, Fab', F(ab')2, scFv, Fv, dsFv диатело и Fd фрагменты. Антительное захватывающее средство можно получать с помощью какого-либо средства. Например, антительное захватывающее средство можно получать ферментативно или химически посредством фрагментирования интактного антитела и/или его можно получать рекомбинатным путем из гена, кодирующего частичную последовательность антитела. Антительное захватывающее средство может содержать одноцепочечный фрагмент антитела. Альтернативно или дополнительно, антительное захватывающее средство может содержать несколько цепей, которые соединены вместе, например, посредством дисульфидных связей; и какие-либо функциональные фрагменты, полученные из таких молекул, где такие фрагменты сохраняют свойства специфического связывания исходной молекулы антитела. В силу своих меньших размеров в виде функциональных компонентов полноразмерной молекулы, фрагменты антител могут давать преимущества над интактными антителами для использования в определенных иммунохимических способах и экспериментальных приложениях.
[00253] Подходящие захватывающие средства, которые можно использовать для практической реализации изобретения, также включают аптамеры. Аптамеры представляют собой олигонуклеотидные последовательности, которые могут связываться со своими мишенями специфически через уникальные трехмерные (3D) структуры. Аптамер может содержать любое подходящее число нуклеотидов, и другие аптамеры могут иметь то же или отличающееся число нуклеотидов. Аптамеры могут представлять собой ДНК или РНК или химически модифицированные нуклеиновые кислоты и могут быть одноцепочечными, двухцепочечными или содержать двухцепочечные области и могут содержать структуры более высокого порядка. Аптамер также может представлять собой фотоаптамер, где фотореакционная или химически реакционная функциональная группа включена в аптамер для того, чтобы сделать возможным его ковалентное соединение с соответствующей ему мишенью. Использование аптамерного захватывающего средства может включать использование двух или больше аптамеров, которые специфически связывают тот же биологический маркер. Аптамер может содержать метку. Аптамер можно идентифицировать с использованием какого-либо известного способа, включая процесс SELEX (систематическая эволюция лигандов экспоненциальным обогащением). После идентификации аптамер можно получать или синтезировать в соответствии с каким-либо известным способом, включая способы химического синтетеза и способы ферментативного синтетеза, и использовать в различных применениях для обнаружения биологического маркера. Liu et al., Curr Med Chem. 18(27):4117-25 (2011). Захватывающие средства, которые можно использовать при практическом осуществлении способов по изобретению, также включают SOMAmer (Slow Off-Rate Modified Aptamers), известные в данной области как обладающие усовершенствованными характеристиками скорости диссоциации. Brody et al., J Mol Biol. 422(5):595-606 (2012). SOMAmer можно создавать с использованием какого-либо известного способа, включая способ SELEX.
[00254] Специалистам в данной области будет ясно, что биологические маркеры можно модифицировать перед анализом для того, чтобы усовершенствовать их разрешение или определять их отличительные черты. Например, биологические маркеры можно подвергать протеолитическому расщеплению перед анализом. Можно использовать любую протеазу. Протеазы, такие как трипсин, которые вероятно расщепляют биологические маркеры на дискретное число фрагментов, являются особенно эффективными. Фрагменты, которые являются результатом расщепления, выполняют функцию сигнатуры для биологических маркеров, тем самым делая возможным их опосредованное обнаружение. В частности, это можно использовать, когда имеют место биологические маркеры со схожими молекулярными массами, что может вносить путаницу с рассматриваемым биологическим маркером. Также протеолитическое фрагментирование можно использовать для высокомолекулярных биологических маркеров, поскольку биологические маркеры меньшего размера намного легче разрешать посредством масс-спектрометрии. В другом примере биологические маркеры можно модифицировать для усовершенствования разрешения обнаружения. Например, нейраминидазу можно использовать для того, чтобы удалять конецвые остатки сиаловой кислоты из гликопротеинов, чтобы усовершенствовать связывание с анионным адсорбентом и усовершенствовать разрешение обнаружения. В другом примере биологическые маркеры можно модифицировать посредством прикрепления метки с конкретной молекулярной массой, которая специфически связывается с молекулярными биологическими маркерами, что дополнительно отличает их. Необязательно, после обнаружения таких модифицированных биологических маркеров отличительные черты биологических маркеров дополнительно можно определять посредством сопоставления физических и химических характеристик модифицированных биологических маркеров в базе данных о белках (например, SwissProt).
[00255] Кроме того, в данной области ценно, что биологические маркеры в образце можно захватывать на подложке для обнаружения. Традиционные подложки включают покрытые антителами 96-луночные планшеты или нитроцеллюлозные мембраны, которые впоследствии исследуют на присутствие белков. Альтернативно, связывающие белок молекулы, прикрепленные к микросферам, микрочастицам, микробусам, бусам или другим частицам, можно использовать для захвата и обнаружения биологических маркеров. Связывающие белок молекулы могут представлять собой антитела, пептиды, пептоиды, аптамеры, низкомолекулярные лиганды или другие связывающие белок захватывающие средства, прикрепленные к поверхности частиц. Каждая связывающая белок молекула может содержать уникальную поддающуюся обнаружению метку, которую кодируют так, что ее можно отличать от других поддающихся обнаружению меток, прикрепленных к другим связывающим белок молекулам для того, чтобы сделать возможным обнаружение биологических маркеров в мультиплексных анализах. Примеры включают, но не ограничиваясь этим, кодированные цветом микросферы с известными интенсивностями флуоресцентного света (см., например, микросферы с технологией xMAP, которые производит Luminex (Austin, Tex.); микросферы, содержащие нанокристаллы квантовых точек, например, имеющие различные соотношения и комбинации цветов квантовых точек (например, нанокристаллы Qdot, которые производит Life Technologies (Carlsbad, Calif.); стекло, покрытое металлическими наночастицами (см., например, нанометки SERS, которые производит Nanoplex Technologies, Inc. (Mountain View, Calif.); материалы со штриховыми кодами (см., например, исчерченные металлические стержни субмикронных размеров, такие как Nanobarcode, которые производит Nanoplex Technologies, Inc.), кодированные микрочастицы с цветными штриховыми кодами (см., например, CellCard, которые производит Vitra Bioscience, vitrabio.com), стеклянные микрочастицы с цифровыми голографическими кодовыми изображениями (см., например, микробусы CyVera, которые производит Illumina (San Diego, Calif.); хемилюминесцентные красители, комбинации соединений красителей; и бусы различных размеров, поддающихся обнаружению.
[00256] В другом аспекте биочипы можно использовать для захвата и обнаружения биологических маркеров по изобретению. Многие белковые биочипы известны в данной области. Они включают, например, белковые биочипы, которые производит Packard BioScience Company (Meriden Conn.), Zyomyx (Hayward, Calif.) и Phylos (Lexington, Mass.). В целом, белковые биочипы содержат подложку, имеющую определенную поверхность. Захватывающий реактив или адсорбент прикрепляют к поверхности подложки. Часто поверхность содержит множество местоположений, с которыми можно адресно взаимодествовать, каждое из этих местоположений имеет захватывающее средство, связанное с ним. Захватывающее средство может представлять собой биологическую молекулу, такую как полипептид или нуклеиновая кислота, которая захватывает другие биологические маркеры конкретным образом. Альтернативно, захватывающее средство может представлять собой хроматографический материал, такой как анионообменный материал, или гидрофильный материал. Примеры белковых биочипов хорошо известны в данной области.
[00257] Настоящее раскрытие также предусматривает способы предсказания вероятности преждевременных родов, которые включают измерение изменения значения обращения пары биологических маркеров. Например, биологический образец можно приводить в контакт с панелью, содержащей один или несколько связывающих полинуклеотид средств. Затем экспрессию одного или нескольких обнаруживаемых биологических маркеров можно оценивать в соответствии со способами, раскрытыми далее, например, с использованием или без использования способов амплификации нуклеиновых кислот. Квалифицированые практики примут во внимание, что в способах, описанных в настоящем описании, измерение экспрессии гена можно автоматизировать. Например, можно использовать систему, которая может осуществлять мультиплексное измерение экспрессии гена, например, предоставляя цифровые показания относительного содержания сотен видов мРНК одновременно.
[00258] В некоторых вариантах осуществления способы амплификации нуклеиновых кислот можно использовать для того, чтобы обнаруживать полинуклеотидный биологический маркер. Например, при амплификации можно использовать олигонуклеотидные праймеры и зонды по настоящему изобретению и способы обнаружения, в которых используют субстраты нуклеиновых кислот, выделенные какими-либо из множества общеизвестных и общепринятых способов (например, Sambrook et al., Molecular Cloning, A laboratory Manual, стр. 7.37-7.57 (2-е изд., 1989); Lin et al., в Diagnostic Molecular Microbiology, Principles and Applications, стр. 605-16 (ред. Persing et al. (1993); Ausubel et al., Current Protocols in Molecular Biology (2001 и последующие обновления)). Способы амплификации нуклеиновых кислот включают, но не ограничиваясь этим, например, полимеразную цепную реакцию (ПЦР) и ПЦР с обратной транскрипцией (RT-PCR) (см., например, патенты США №№ 4683195; 4683202; 4800159; 4965188), лигазную цепную реакцию (LCR) (см., например, Weiss, Science 254:1292-93 (1991)), амплификацию замещением цепей (SDA) (см. например, Walker et al., Proc. Natl. Acad. Sci. USA 89:392-396 (1992); патенты США №№ 5270184 и 5455166), термофильную SDA (tSDA) (см., например, европейский патент № 0 684 315) и способы, описанные в патенте США № 5130238; Lizardi et al., BioTechnol. 6:1197-1202 (1988); Kwoh et al., Proc. Natl. Acad. Sci. USA 86:1173-77 (1989); Guatelli et al., Proc. Natl. Acad. Sci. USA 87:1874-78 (1990); патентах США №№ 5480784; 5399491; публикации США № 2006/46265.
[00259] В некоторых вариантах осуществления измерение мРНК в биологическом образце можно использовать в качестве суррогата для обнаружения уровня соответствующего белкового биологического маркера в биологическом образце. Таким образом, какие-либо из биологических маркеров, пар биологических маркеров или панелей обращения биологических маркеров, описанных в настоящем описании, также можно обнаруживать посредством обнаружения подходящей РНК. Уровни мРНК можно измерять посредством количественной полимеразной цепной реакции с обратной транскрипцией (RT-PCR с последующей qPCR). RT-PCR используют для того, чтобы создавать кДНК из мРНК. кДНК можно использовать в qPCR анализе для создания флуоресценции по мере продвижения процесса амплификации ДНК. Посредством сравнения с калибровочной кривой, qPCR позволяет получать абсолютное измерение, такое как число копий мРНК на клетку. Нозерн-блоттинг, микрочипы, анализы Invader и RT-PCR в комбинации с капиллярным электрофорезом используют для того, чтобы измерять уровни экспрессии мРНК в образце. См. Gene Expression Profiling: Methods and Protocols, ред. Richard A. Shimkets, Humana Press, 2004.
[00260] Некоторые варианты осуществления, описанные в настоящем описании, относятся к диагностическим и прогностическим способам определения вероятности преждевременных родов у беременной женщины. Обнаружение уровня экспрессии одного или нескольких биологических маркеров и/или определение соотношения биологических маркеров можно использовать для того, чтобы определять вероятность преждевременных родов у беременной женщины. Такие способы обнаружения можно использовать, например, для ранней диагностики состояния, чтобы определять, предрасположен ли субъект к преждевременным родам, чтобы осуществлять мониторинг прогресса преждевременных родов или прогресса протоколов лечения, чтобы оценивать тяжесть преждевременных родов, чтобы предсказывать исход преждевременных родов и/или перспективу извлечения или родов в полный срок или чтобы содействовать определению подходящего лечения для преждевременных родов.
[00261] Количественное определение биологических маркеров в биологическом образце можно осуществлять, без ограничения, посредством способов, описанных выше, а также какого-либо другого способа, известного в данной области. Количественные данны, полученные таким образом, затем подвергают процессу аналитической классификации. В таком процессе осуществляют манипуляции с исходными данными в соответствии с алгоритмом, где алгоритм предварительно определен с помощью обучающего набора данных, например, как описано в примерах, приведенных в настоящем описании. Алгоритм может использовать обучающий набор данных, предоставленный в настоящем описании, или может использовать руководства, предоставленные в настоящем описании, чтобы создавать алгоритм с использованием другого набора данных.
[00262] В некоторых вариантах осуществления анализ поддающейся измерении особенность для того, чтобы определять вероятность преждевременных родов у беременной женщины, включает использование предсказательной модели. В дополнительных вариантах осуществления анализ поддающейся измерению особенности для того, чтобы определять вероятность преждевременных родов у беременной женщины, включает сравнения указанной поддающейся измерению особенности с эталонной особенностью. Как признают специалисты в данной области, такое сравнение может представлять собой непосредственное сравнение с эталонной особенностью или опосредованное сравнение, при котором эталонная особенность встроена в предсказательную модель. В дополнительных вариантах осуществления анализ поддающейся измерению особенности для того, чтобы определять вероятность преждевременных родов у беременной женщины, включает одно или несколько из модели линейного дискриминантного анализа, алгоритма классификации способом опорных векторов, модели рекурсивной элиминации признаков, модели прогностического анализа микрочипа, логистической регрессионной модели, алгоритма CART, алгоритма FlexTree, алгоритма LART, алгоритма случайного леса, алгоритма MART, алгоритма машинного обучения, способа регрессии со штрафом или их сочетания. В конкретных вариантах осуществления анализ включает логистическую регрессию.
[00263] В процессе аналитической классификации можно использовать какой-либо один из множества статистических аналитических способов для осуществления манипуляций с количественными данными и обеспечения классификации образца. Примеры эффективных способов включают линейный дискриминантный анализ, рекурсивную элиминацию признаков, прогностический анализ микрочипа, логистическую регрессию, алгоритм CART, алгоритм FlexTree, алгоритм LART, алгоритм случайного леса, алгоритм MART, алгоритмы машинного обучения; и т. д.
[00264] Для создания случайного леса для предсказания GAB специалист в данной области может рассматривать набор из k субъектов (беременных женщин), для которых известен гестационный возраст при рождении (GAB) и для которых N анализируемых веществ (переходов) измерены в образце крови, взятом за несколько недель до рождения. Дерево регрессии начинается с корневого узла, который содержит все субъекты. В корневом узле можно вычислять усредненный GAB для всех субъектов. Дисперсия GAB в корневом узле будет высокой, поскольку в нем присутствует смесь женщин с различными GAB. Затем корневой узел делят (подразделяют) на две ветви с тем, чтобы каждая ветвь содержала женщин со схожими GAB. Снова вычисляют усредненный GAB для субъектов на каждой ветви. Дисперсия GAB на каждой ветви будет ниже, чем в корневом узле, поскольку поднабор женщин на каждой ветви имеет относительно более схожие GAB, чем те, которые находятся в корневом узле. Создают две ветви посредством выбора анализируемого вещества и порогового значения для анализируемого вещества, что создает ветви со схожими GAB. Анализируемое вещество и пороговое значение выбирают среди набора из всех анализируемых веществ и пороговых значений, обычно со случайным поднабором анализируемых веществ в каждом узле. Процедуру продолжают рекурсивно, создавая ветви для того, чтобы создавать листья (конечные узлы), в которых субъекты имеют очень схожие GAB. Предсказанный GAB в каждом концевом узле представляет собой усредненный GAB для субъектов в этом концевом узле. Эта процедура создает одно дерево регрессии. Случайный лес может состоять из нескольких сотен или нескольких тысяч таких деревьев.
[00265] Классификацию можно выполнять в соответствии со способами предсказательного моделирования, которые задают порог для определения вероятности того, что образец принадлежит к данному классу. Вероятность предпочтительно составляет по меньшей мере 50%, или по меньшей мере 60%, или по меньшей мере 70%, или по меньшей мере 80% или выше. Классификацию также можно выполнять посредством определения того, дает ли сравнение между полученным массивом данных и эталонным массивом данных статистически значимое различие. Если так, то образец, из которого получали массив данных, классифицируют как не принадлежащий классу эталонного массива данных. Наоборот, если такое сравнение не дает статистически значимое различие с эталонным массивом данных, то образец, из которого получали массив данных, классифицируют как относящийся к классу эталонного массива данных.
[00266] Предсказательную способность модели можно оценивать в соответствии с ее способностью предоставлять метрику качества, например, AUROC (площадь под ROC-кривой), или точность конкретного значения или диапазона значений. Меры площади под кривой можно использовать для сравнения точности классификатора во всем диапазоне данных. Классификаторы с большей AUC имеют более высокую способность классифицировать неизвестное правильно между двумя группами, представляющими интерес. В некоторых вариантах осуществления желаемый порог качества представляет собой предсказательную модель, которая классифицирует образец с точностью по меньшей мере приблизительно 0,5, по меньшей мере приблизительно 0,55, по меньшей мере приблизительно 0,6, по меньшей мере приблизительно 0,7, по меньшей мере приблизительно 0,75, по меньшей мере приблизительно 0,8, по меньшей мере приблизительно 0,85, по меньшей мере приблизительно 0,9, по меньшей мере приблизительно 0,95 или выше. В качестве альтернативной меры желаемый порог качества может относиться к предсказательной модели, которая будет классифицировать образец с AUC по меньшей мере приблизительно 0,7, по меньшей мере приблизительно 0,75, по меньшей мере приблизительно 0,8, по меньшей мере приблизительно 0,85, по меньшей мере приблизительно 0,9 или выше.
[00267] Как известно в данной области, относительную чувствительность и специфичность предсказательной модели можно корректировать для того, чтобы способствовтаь или метрике избирательности или метрике чувствительности, где две метрики имеют обратную зависимость. Пределы в модели, как описано выше, можно корректировать для обеспечения выбранного уровня чувствительности или специфичности, в зависимости от конкретных требований выполняемого теста. Чувствительность и/или специфичность может составлять по меньшей мере приблизительно 0,7, по меньшей мере приблизительно 0,75, по меньшей мере приблизительно 0,8, по меньшей мере приблизительно 0,85, по меньшей мере приблизительно 0,9 или выше.
[00268] Исходные данные можно изначально анализировать посредством измерения значений для каждого биологического маркера, обычно в трех повторениях или несколько раз в трех повторениях. Можно осуществлять манипуляции с данными можно, например, исходные данные можно преобразовывать с использованием калибровочных кривых и использовать усредненное трех измерений для того, чтобы вычислять усредненное и стандартное отклонение для каждого пациента. Эти значения можно преобразовывать перед использованием в моделях, например, проводить логарифмическое преобразование, преобразование Бокса-Кокса (Box and Cox, Royal Stat. Soc., Series B, 26:211-246(1964). Затем данные вводят в предсказательную модель, которая классифицирует образец в соответствии с состоянием. Результрующую информацию можно передавать пациенту или поставщику медицинских услуг.
[00269] Для того чтобы создавать предсказательную модель для преждевременных родов, в обучающем наборе используют надежный набор данных, который содержит известные контрольные образцы и образцы, соответствующие классификации преждевременных родов, представляющей интерес. Размер образца можно выбирать с использованием общепринятых критериев. Как рассмотрено выше, различные статистические способы можно использовать для получения высокоточной предсказательной модели. Примеры такого анализа приведены в примере 2.
[00270] В одном из вариантов осуществления иерархическую кластеризацию осуществляют при получении предсказательной модели, где корреляцию Пирсона используют в качестве метрики кластеризации. Один подход состоит в том, чтобы рассмотреть массив данных преждевременных родов в качестве «обучающего образца» в проблеме «обучения с учителем». CART представляет собой стандарт в медицинских применениях (Singer, Recursive Partitioning in the Health Sciences, Springer (1999)), и его можно модифицировать посредством преобразования каких-либо качественных особенностей в количественные особенности; их сортировки по достигаемым уровням значимости, оцениваемым с помощью способов повторного использования образцов для T2 статистики Хотеллинга; и подходящего применения способа лассо. Проблемы в предсказании превращают в проблемы в регрессии, не теряя из виду предсказание, в действительности, делая подходящим использование критерия Джинни для классификации при оценке качества регрессии.
[00271] Этот подход привел к тому, что называют FlexTree (Huang, Proc. Nat. Acad. Sci. U.S.A 101:10529-10534(2004)). FlexTree работает очень хорошо при симуляции и в применении к многим формам данных, и его можно использовать для практической реализации заявленных способов. Разработано программное обеспечение, автоматизирующее FlexTree. Альтернативно, можно использовать LARTree или LART (Turnbull (2005) Classification Trees with Subset Analysis Selection by the Lasso, Stanford University). В названии отражены бинарные деревья, как в CART и FlexTree; лассо, как отмечено; и реализация лассо через то, что названо LARS авторами Efron et al. (2004) Annals of Statistics 32:407-451 (2004). Также см. Huang et al., Proc. Natl. Acad. Sci. USA. 101(29):10529-34 (2004). Другие способы анализа, которые можно использовать, включают логическую регрессию. Один способ логической регрессии Ruczinski, Journal of Computational and Graphical Statistics 12:475-512 (2003). Логическая регрессия походит на CART в том отношении, что ее классификатор можно отображать как бинарное дерево. Она отличается тем, что каждый узел имеет булевы выражения относительно особенностей, которые являются более общими, чем простые выражения «и», создаваемые с помощью CART.
[00272] Другой подход заключается в ближайших сжатых центроидах (Tibshirani, Proc. Natl. Acad. Sci. U.S.A 99:6567-72(2002)). Технология похожа на k-средние, но имеет такое преимущество, что посредством сжатия центров кластеров автоматически выбирают особенности, как в случае с лассо, чтобы фокусировать внимание на небольшом числе тех, которые информативны. Подход доступен в виде программного обеспечения PAM и широко используется. Две другие группы алгоритмов, которые можно использовать, представляют собой случайный лес (Breiman, Machine Learning 45:5-32 (2001)) и MART (Hastie, The Elements of Statistical Learning, Springer (2001)). Эти два способа известны в данной области как «способы комитета», в которых используют предикторы, которые «голосуют» за исход.
[00273] Для обеспечения упорядочения по значимости можно определять уровень ложноположительных результатов (FDR). Сначала генерируют набор распределений значений несхожести для нулевой гипотезы. В одном из вариантов осуществления осуществляют перестановку значений наблюдаемых профилей для того, чтобы создавать последовательность распределений коэффициентов корреляции, получаемых для низкой вероятности, тем самым создавая подходящий набор распределений коэффициентов корреляции для нулевой гипотезы (Tusher et al., Proc. Natl. Acad. Sci. U.S.A 98, 5116-21 (2001)). Набор распределения для нулевой гипотезы получают посредством: перестановки значений каждого профиля для всех доступных профилей; вычисления попарных коэффициентов корреляции для всех профилей; вычисления функции плотности вероятности для коэффициентов корреляции для этой перестановки; и повторения процедуры N раз, где N представляет собой большое число, обычно 300. Используя N распределений, вычисляют подходящую меру (среднее, медиану и т. д.) счета значений коэффициентов корреляции, чтобы их значения превышали значение (сходства), которое получают из распределения экспериментально наблюдаемых значений сходства при заданном уровне значимости.
[00274] FDR представляет собой соотношение числа ожидаемых ложных значимых корреляций (которое оценивают по корреляциям больше выбранной корреляции Пирсона в наборе случайных данных) и числа корреляций больше выбранной корреляции Пирсона в эмпирических данных (значимые корреляции). Это значение корреляции порога можно применять к корреляциям между экспериментальными профилями. Используя указанное выше распределение, для значимости выбирают уровень доверия. Это используют для того, чтобы определять наименьшее значение коэффициента корреляции, которое превышает результат, который будет получен случайным образом. Используя этот способ, получают пороги для положительной корреляции, отрицательной корреляции или обеих. Используя этот порог(и), пользователь может фильтровать наблюдаемые значения попарных коэффициентов корреляции и устранять те, которые не превышают порог(и). Кроме того, оценку уровня ложноположительных можно получать для заданного порога. Для каждого из индивидуальных распределений «случайной корреляции» можно находить, сколько наблюдений попадают за пределы порогового диапазона. Эта процедура предусматривает последовательность счетов. Среднее и стандартное отклонение последовательности предоставляют усредненное число потенциальных ложноположительных и их стандартное отклонение.
[00275] В альтернативном аналитическом подходе переменные, выбранные в кросс-секционном анализе, отдельно используют в качестве предикторов в анализе времени до события (анализе выживаемости), где событие представляет собой наступление преждевременных родов, и субъектов без события считают цензурированными во время родов. Учитывая конкретный исход беременности (событие преждевременных родов или отсутствие события), случайные длительности времени, в течение которого наблюдают каждого пациента, и отбор протеомных и других особенностей, параметрический подход к анализу выживаемости может быть лучше, чем широко применяемая полупараметрическая модель Кокса. Параметрическая аппроксимация выживаемости Вейбулла допускает монотонное увеличение, снижение или постоянный уровень опасности, и также имеет пропорциональное представление опасности (как это сделано в модели Кокса) и представление времени до ускоренного отказа. Все стандартные инструменты, доступные при получении приблизительных оценок максимальной вероятности для коэффициентов регрессии и соответствующих функций, доступны при использовании этой модели.
[00276] Вдобавок модели Кокса можно использовать, в частности, поскольку снижение числа ковариат до размера, удобного в обращении, с использованием лассо значительно упрощает анализ, делая возможным непараметрический или полупараметрический подход к предсказанию времени до преждевременных родов. Эти статистические инструменты известны в данной области и применимы ко всем типам протеомных данных. Предусмотрен набор данных о биологических маркерах, клинических и генетических данных, которые можно легко определять и которые высоко информативны в отношении вероятности преждевременных родов и предсказанного времени до события преждевременных родов у указанной беременной женщины. Также алгоритмы предоставляют информацию относительно вероятности преждевременных родов у беременной женщины.
[00277] Соответственно, специалист в данной области понимает, что вероятность преждевременных родов в соответствии с изобретением можно определять, используя количественную или категориальную переменную. Например, при практическом осуществлении способов по изобретению поддающуюся измерению особенность каждого из N биологических маркеров можно подвергать категориальному анализу данных для того, чтобы определять вероятность преждевременных родов как бинарный категориальный исход. Альтернативно, способы по изобретению позволяют анализировать поддающуюся измерению особенность каждого из N биологических маркеров посредством изначального вычисления количественных переменных, в частности, предсказанного гестационного возраста при рождении. Предсказанный гестационный возраст при рождении впоследствии можно использовать в качестве основы для того, чтобы предсказать риск преждевременных родов. Изначально используя количественную переменную и впоследствии преобразуя количественную переменную в категориальную переменную, способы по изобретению учитывают непрерывность измерений, обнаруживаемых для поддающихся измерению особенностей. Например, посредством предсказания гестационного возраста при рождении вместо выполнения бинарного предсказания преждевременных родов в сравнении с полносрочными родами, возможно адаптировать лечение для беременной женщины. Например, более ранний предсказанный гестационный возраст при рождении будет вести к более интенсивному пренатальному вмешательству, т. е. мониторингу и лечению, чем предсказанный гестационный возраст, который приближается к полному сроку.
[00278] Среди женщин с предсказанным GAB в j суток±k суток, p(PTB) можно оценивать как долю женщин в клиническом исследовании PAPR (см. пример 1) с предсказанным GAB в j суток±k суток, которые фактически рожают до 37 недель гестационного возраста. В более общем смысле, для женщин с предсказанным GAB в j суток±k суток, вероятность того, что фактический гестационный возраст при рождении будет меньше, чем конкретный гестационный возраст, p(фактический GAB < конкретный GAB), оценивали как долю женщин в клиническом исследовании PAPR с предсказанным GAB в j суток±k суток, которые фактически рожают прежде конкретного гестационного возраста.
[00279] При разработке предсказательной модели может быть желательно выбирать поднабор маркеров, т. е. по меньшей мере 3, по меньшей мере 4, по меньшей мере 5, по меньшей мере 6, вплоть до полного набора маркеров. Обычно выбирают поднабор маркеров, который обеспечивает потребности количественного анализа образца, например, доступность реактивов, удобство количественного определения и т. д., при этом сохраняя высоко точную предсказательную модель. Отбор определенного числа информативных маркеров для построения классификационных моделей требует определения для метрики эффективности и задаваемого пользователем порога для получения модели с эффективной предсказательной способностью на основании этой метрики. Например, метрика эффективности может представлять собой AUC, чувствительность и/или специфичность предсказания, а также общую точность предскательной модели.
[00280] Как поймут специалисты в данной области, в процессе аналитической классификации можно использовать какой-либо один из множества статистических аналитических способов, чтобы осуществлять манипуляции с количественными данными и обеспечивать классификацию образца. Примеры эффективных способов включают, без ограничения, линейный дискриминантный анализ, рекурсивную элиминацию признаков, прогностический анализ микрочипа, логистическую регрессию, алгоритм CART, алгоритм FlexTree, алгоритм LART, алгоритм случайного леса, алгоритм MART и алгоритмы машинного обучения. При обучении модели используют различные способы. Выбор поднабора маркеров может быть предназначен для прямого выбора или пошагового исключения из поднабора маркеров. Можно выбирать число маркеров, которе будет оптимальным для эффективности модели без использования всех маркеров. Один путь, чтобы определить оптимальное число членов, состоит в выборе числа членов, которое позволяет создавать модель с желаемой предсказательной способностью (например, AUC>0,75 или эквивалентные меры чувствительности/специфичности), которая лжет в пределах не больше одной стандартной ошибки от максимального значения, получаемого для этой метрики, используя какую-либо комбинацию и число членов, используемых для данного алгоритма.
[00281] В еще одном аспекте изобретение относится к комплектам для определения вероятности преждевременных родов. Комплект может включать одно или несколько средств для обнаружения биологических маркеров, контейнер для размещения биологического образца, выделенного у беременной женщины; и печатные инструкции для средств, взаимодействующих с биологическим образцом или частью биологического образца, чтобы обнаруживать присутствие или количество выделенных биологических маркеров в биологическом образце. Средства можно упаковывать в отдельные контейнеры. Комплект дополнительно может содержать один или несколько контрольных эталонных образцов и реактивов для осуществления иммунологического анализа.
[00282] Комплект может содержать один или несколько контейнеров для композиций, содержащихся в комплекте. Композиции могут быть в жидкой форме или могут быть лиофилизированными. Подходящие контейнеры для композиций включают, например, бутылки, флаконы, шприцы и пробирки. Контейнеры можно формировать из различных материалов, включая стекло или пластмассу. Комплект также может содержать вкладыш в упаковку, содержащий письменные инструкции для способов определения вероятности преждевременных родов.
[00283] Из приведенного выше описания очевидно, что вариации и модификации можно выполнять в изобретении, описанном в настоящем описании, чтобы адаптировать его к различным использованиям и условиям. Такие варианты осуществления также входят в объем следующей формулы изобретения.
[00284] Изложение списка элементов в каком-либо определении для переменной в настоящем описании включает определения для этой переменной в виде любого отдельного элемента или комбинации (или подкомбинации) перечисленных элементов. Изложение варианта осуществления в настоящем описании включает этот вариант осуществления в виде любого отдельного варианта осуществления или в комбинации с какими-либо другими вариантами осуществления или их частями.
[00285] Все патенты и публикации, упомянутые в этом описании, включены в настоящее описание посредством ссылки в той же степени, как если бы каждый независимый патент и публикация были конкретно и индивидуально отмечены как включенные по ссылке.
[00286] Следующие примеры предоставлены в качестве иллюстрации, но не ограничения.
ПРИМЕРЫ
Пример 1. Разработка набора образцов для исследования и валидации биологических маркеров для преждевременных родов
[00287] Разрабатывали стандартный протокол для проведения клинического исследования протеомной оценки преждевременного риска (PAPR). Образцы получали у женщин в 11 местах, одобренных Institutional Review Board (IRB), по всем Соединенным Штатам. После предоставления информированного согласия получали образцы сыворотки и плазмы, а также уместную информацию, касающуюся демографических характеристик пациента, прошлого медицинского анамнеза и анамнеза прошлой беременности, анамнеза текущей беременности и сопутствующих лекарственных средств. После родов собирали данные, касающиеся состояния матери и младенца и осложнений. Образцы сыворотки и плазмы обрабатывали в соответствии с протоколом, который требует стандартизованного охлажденного центрифугирования, разделения образцов на аликвоты в криофлаконы с 2D штриховыми кодами и последующей заморозки при -80°C.
[00288] После родов, случаи преждевременных родов индивидуально рассматривали для того, чтобы определять их статус в качестве самопроизвольных преждевременных родов или медицински показанных преждевременных родов. Для этого анализа использовали только случаи самопроизвольных преждевременных родов. Для исследования биологических маркеров преждевременных родов анализировали образцы сыворотки для 86 преждевременных случаев и 172 контролей, покрывающие гестационный возраст при взятии крови (GABD) от 17 недель и 0 суток (17,0) до 28 недель и 6 суток (28,6). В целях верификации также анализировали отдельный набор образцов, который состоял из сыворотки из 50 преждевременных случаев и 100 контролей, для того же диапазона гестационного возраста. Для каждого случая по GABD сопоставляли два контроля, которые выбирали из нескольких случайно сгенерированных панелей контролей, которые отвечали распределению родов, приведенному в 2012 National Vital Statistics Report. Протокол создан так, чтобы гарантировать, что сотрудникам лаборатории не известен гестационный возраст при рождении и статус случая или контроля у субъектов, использованных для обоих наборов образцов. Сотрудники, отвечающие за информатику, также не имели сведений о верификационном наборе образцов до завершения аналитического анализа образцов.
[00289] Образцы сыворотки истощали по белкам с высоким относительным содержанием, используя Human 14 Multiple Affinity Removal System (MARS 14), которая удаляет 14 наиболее обильных белков, которые обрабатывают как неинформативные в отношении идентификации изменений, релевантных для заболевания, в сывороточном протеоме. С этой целью равные объемы (50 мкл) каждого клинического, объединенного образца сыворотки человека (HGS) или объединенного образца сыворотки беременной женщины (pHGS) разводили в 150 мкл буфера A для колонки Agilent и фильтровали в фильтровальных планшетах Captiva для того, чтобы удалять преципитаты. Фильтрованные образцы истощали с использованием колонки MARS-14 (4,6×100 мм, № по каталогу 5188-6558, Agilent Technologies), в соответствии с протоколом производителя. Образцы охлаждали до 4°C в автоматическом пробоотборнике, колонку для истощения прогоняли при комнатной температуре и собранные фракции хранили при 4°C до дальнейшего анализа. Несвязанные фракции собирали для дальнейшего анализа.
[00290] Истощенные образцы сыворотки восстанавливали дитиотреитолом, алкилировали с использованием йодацетамида и затем расщепляли с использованием 5,0 мкг Trypsin Gold - Mass Spec Grade (Promega) при 37°C в течение 17 часов (±1 час). После расщепления трипсином, смесь 187 пептидов Stable Isotope Standard (SIS) добавляли в образцы и половину каждого образца обессоливали на Empore C18 96-well Solid Phase Extraction Plate (3M Bioanalytical Technologies). Планшет кондиционировали в соответствии с протоколом изготовителя. Пептиды промывали с использованием 300 мкл 1,5% трифторуксусной кислоты, 2% ацетонитрила, элюировали с использованием 250 мкл 1,5% трифторуксусной кислоты, 95% ацетонитрила, замораживали при -80°C в течение 30 минут и затем лиофилизировали досуха. Лиофилизированные пептиды восстанавливали в 2% ацетонтиле/0,1% муравьиной кислоте, содержащих три не относящихся к человеку внутренних стандартных (IS) пептида. Пептиды разделяли в 30 мин градиенте ацетонитрила при 400 мкл/мин на колонке Agilent Poroshell 120 EC-C18 (2,1×100 мм, 2,7 мкм) при 40°C и впрыскивали в масс-спектрометр Agilent 6490 Triple Quadrapole.
[00291] Истощенные и расщепленные трипсином образцы анализировали с использованием способа запланированного мониторинга нескольких реакций (sMRM). В sMRM анализе осуществляли мониторинг 898 переходов, в которых измеряли 259 биологических пептидов и 190 IS пептидов (187 SIS+3 IS), которые представляли 148 белков. Хроматографические пики интегрировали с использованием программного обеспечения Mass Hunter Quantitative Analysis (Agilent Technologies).
[00292] АНАЛИЗ ДАННЫХ
[00293] Анализ данных исследования и верификационных образцов осуществляли в две фазы. В первой фазе надежные биологические маркеры идентифицировали посредством отбора с использованием образцов исследования и подтверждения с использованием независимого набора верификационных образцов. Во второй фазе данные исследования и верификации комбинировали и использовали для того, чтобы идентифицировать наилучшие анализируемые вещества и панели анализируемых веществ для разработки классификатора.
[00294] Фаза I: слепой анализ
[00295] Начальную разработку классификатора сосредоточили на гестационном возрасте от 17,0 до 25,6. Используя образцы исследования, набор пептидов, соответствующих 62 белкам, отбирали на основании преданалитических и аналитических критериев. Диагностическую эффективность анализируемых веществ оценивали в серии узких интервалов GABD, которые покрывали три недели с перекрытием в две недели между смежными интервалами. На основании согласованности диагностической эффективности (повышающая и понижающая регуляция в случаях в сравнении с контролями по всему GABD), поднабор из 43 анализируемых веществ выбирали для дальнейшего анализа.
[00296] Для каждого узкого GABD интервала формировали набор обращений с использованием всех комбинаций анализируемых веществ под повышающей и понижающей регуляцией в узком интервале. Значение обращения представляет собой соотношение относительной площади пика анализируемого вещества под повышающей регуляцией и относительной площади пика анализируемого вещества под понижающей регуляцией, и оно служит как для нормализации вариабельности, так и усиления диагностического сигнала. Из всех возможных обращений в узком интервале выбирали поднабор на основании их индивидуальной одномерной эффективности (AUC≥0,6).
[00297] Для каждого интервала формировали панели обращения различных размеров (размеры 2, 3, 4, 6, 8). Для каждого размера панели в интервале осуществляли перекрестную валидацию способом Монте-Карло (MCCV) посредством обучения и тестирования логистического классификатора итерационно 1000 раз на 70% и 30% образцов, соответственно. Размер панели 4, который определяли как оптимальный по средней MCCV AUC, впоследствии использовали для идентификации кандидатных обращений, которые хорошо работали на панелях. Кандидатные обращения идентифицировали по частоте встречаемости среди наиболее эффективных логистических классификаторов на панелях размера 4 в MCCV анализе. Для каждого интервала создавали три набора таблиц частот обращения с использованием мер эффективности для AUC или частичной AUC (pAUC) для чувствительности в диапазоне от 0,7 до 1 или корреляции оценки выходных данных классификатора со значением времени до родов (TTB) (разность в сутках между GABD и гестационным возрастом при рождении). Из каждого из этих списоков обращений лучшие 15 обращений отбирали для дальнейшего анализа.
[00298] Для каждого узкого интервала GABD панели обращения размера 2, 3, 4 формировали из каждого из трех списков (AUC, pAUC и TTB) и на основании эффективности MCCV анализа отбирали лучшие 15 панелей для каждого размера панели в каждом интервале. Наряду с лучшими 15 обращениями из каждого из трех списков (AUC, pAUC и TTB) для каждого интервала эти лучшие 15 панелей размера 2, 3, 4 использовали для обучения логистических классификаторов на образцах исследования и генерировали оценки классификации для верификационных образцов слепым образом.
[00299] Сторонний статистик оценивал эффективность всех обращений и панели классификаторов, и приведены AUC, pAUC для ROC-кривых и TTB корреляция оценок классификатора.
[00300] Фаза II: открытый анализ
[00301] После выведения из слепого метода, набор данных исследования и верификации комбинировали и повторно анализировали. Поскольку экспрессия диагностических белков может меняться на протяжении беременности, авторы изобретения исследовали уровни белков в качестве функции GABD. Для создания кинетических графиков применяли медианный сглаживающий интервал±10 суток. Относительные уровни белков выражали как соотношение площади пика эндогенного пептида и соответствующего ему SIS стандарта (удельное соотношение). Примеры белков с уровнями, которые возрастают при беременности, но не различаются в случаях PTB и контролях, представлены на фиг. 3, 4 и 10. Измерение уровней таких белков может быть полезно при точном датировании беременности (например, «хронология» беременности). По хронологии беременности предсказывают гестационный возраст на основании относительного содержания одного или нескольких белков (переходов). Альтернативно, в том же анализе авторы изобретения идентифицировали белки, уровни которых меняются с течением GABD, но демонстрируют различия между случаями PTB и контролями фиг. 5. Эти белки являются очевидными диагностическими кандидатами на разработку классификатора PTB. Влияние формирования обращения, используя соотношение сверхэкспрессированного белка и недоэкспрессированного белка, также показано на примере (фиг. 8 и 21). Ясно, что это ведет к увеличению разделения в случаях PTB и контролях. Предыдущий анализ подсказывал, что на уровни некоторых анализируемых веществ может оказывать влияние индекс массы тела (BMI) до беременности. CLIN. CHEM. 37/5, 667-672 (1991); European Journal of Endocrinology (2004) 150 161-171. По этой причине исследовали влияние BMI на разделение, выражая значение обращения на протяжении беременности только у тех пациентов, BMI которых составляет меньше 35 (фиг. 21). Это ведет к дополнительному усовершенствованию разделения.
[00302] Отбор обращений и разработка классификатора в комбинированном наборе данных исследования и верификации дублировали более ранние исследования. Авторы изобретения сосредоточились на третьем перекрывающемся интервале GABD (сутки 133-153), чтобы пояснять анализ примером. MCCV анализ осуществляли для того, чтобы идентифицировать кандидатные обращения. Для того чтобы оценивать эффективность панелей, значения обращения комбинировали в простом классификаторе LogSum. Классификатор LogSum присваивает оценку каждому образцу на основании суммы логарифмов значения удельного соотношения каждого обращения для этого образца. Отсутствие коэффициентов в классификаторе этого типа помогает избегать проблем чрезмерной подгонки. Любой специалист в данной области может получить эквивалентный логистический классификатор с использованием тех же анализируемых веществ и общепризнанных способов. Многомерная эффективность панели из трех лучших обращений, сформированной из четырех белков, представлена в виде гистограммы значений AUC, получаемых посредством перекрестной валидации, и на ROC-кривых на фиг. 8. Предыдущий анализ показывал, что на уровни некоторых анализируемых веществ может оказывать влияние индекс массы тела (BMI) до беременности.
[00303] Авторы изобретения определяли белки и/или обращения, проиллюстрированные здесь использованием ITIH4/CSH, которые представляют собой мощные предикторы времени до родов (TTB) (фиг. 10). TTB определяют как разность между GABD и гестационным возрастом при рождении (GAB). Это обладает потенциалом сделать возможным предсказание, или индивидуально или в математической комбинации таких анализируемых веществ, чтобы клинически оценивать TTB (или GAB).
Пример 2. Валидация предиктора sPTB IBP4/SHBG
[00304] Этот пример демонстрирует валидацию предиктора sPTB IBP4/SHBG, идентифицированного в большой программе по материнской сывороточной протеомике, у бессимптомных женщин при ранней беременности.
[00305] Субъекты
[00306] Исследование протеомной оценки преждевременного риска (PAPR) проводили по стандартизованному протоколу в 11 местах, одобренных Institutional Review Board (IRB), по всем Соединенным Штатам (clinicaltrials.gov, идентификатор: NCT01371019). Субъектов вводили в исследование между 17 0/7 и 28 6/7 неделями GA. Датировку устанавливали, используя предварительно определенный протокол менструальной датировки с подтверждением с помощью ранней ультразвуковой биометрии или только ультразвук, чтобы предоставлять наилучший клинический оценочный гестационный возраст. Индекс массы тела (BMI) определяли по росту и массе до беременности, которую сообщали самостоятельно. Исключали беременности с множественными беременностями и с известными или предполагаемыми большими аномалиями плода. Собирали уместную информацию относительно демографических характеристик субъекта, прошлого медицинского анамнеза и анамнеза прошлой беременности, анамнеза текущей беременности и сопутствующих лекарственных средств, которые вводили в электронную форму описания случая. После родов собирали данные об исходах матери и младенца и осложнениях. Все роды объявляли полносрочными (≥37 0/7 недель GA), самопроизвольными преждевременными (включая преждевременный разрыв околоплодных оболочек) или медицински показанными преждевременными родами. Как указано, разногласия проясняли с участием главного исследователя в месте исследования. Выполняли экспертное заключение и данные блокировали до валидационных исследований.
[00307] Сбор образцов
[00308] Материнскую кровь собирали и обрабатывали следующим образом: период свертывания 10 минут при комнатной температуре, после чего следовало незамедлительное охлажденное центрифугирование или размещение на ледяной водяной бане при 4-8°C до центрифугирования. Кровь центрифугировали в пределах 2,5 часа от взятия и 0,5 мл аликвоты сыворотки хранили при -80°C до анализа.
[00309] Принципы разработки предиктора
[00310] Разработка предиктора IBP4/SHBG включала независимые и последовательные стадии исследования, верификации и валидации в соответствии с руководствами Institute of Medicine (IOM) по передовому опыту в «-омных» исследованиях. IOM (Institute of Medicine). Evolution of Translation Omics: Lessons Learned and the Path Forward. (Micheel CM, Nass SJ, Omenn GS, eds.). Washington, DC: The National Academies Press.; 2012:1-355. Аналитическая валидация предшествовала клиническому анализу валидационного образца и включала оценку воспроизводимости между партиями и внутри партии, переноса и предела обнаружения.
[00311] Валидационный комбинированный анализ случая/контроля осуществляли на предварительно заданных случаях sPTB и контрольных образцах, независимо от исследования и верификации. Случаи sPTB содержали образцы всего из 9 мест, причем два места уникальны для валидации. Валидационные случаи и контроли проходили 100% локальную верификацию исходных документов с медицинской картой каждого субъекта перед масс-спектрометрическим (MS) анализом сыворотки. Этот процесс гарантировал, что все субъекты отвечают критериям включения и исключения, а также подтверждал медицинские/гестационные осложнения и присвоенный GA при родах для всех субъектов в момент сбора образца и родов. Подробные протоколы анализа, включая план валидационного исследования, план анализа и протокол заслепления, были созданы предвариетльно. Персонал вслепую работал с присвоением данных о случае субъекта, контроле и GA при родах, за исключением директора по клиническим работам (DCO) и менеджера клинических данных. План анализа данных включал предварительно заданные валидационные требования и протокол для двойного независимого внешнего анализа. Оценки предиктора, вычисленные, как описано ниже, определяли для всех образцов субъекта с помощью статистика, работающего вслепую. Данные о случае, контроле и GA, связанные с оценками предиктора директором по клиническим работам DCO, подвергали независимому внешнему статистическому анализу. Затем площадь под кривой рабочей характеристики приемника (AUROC) и результаты тестирования значимости перадавали обратно DCO. Передача данных включала использование функции SUMPRODUCT (Microsoft. Microsoft Excel. 2013), чтобы гарантировать сохранение целостности данных. Чтобы предоставлять контрольный журнал данных от каждого субъекта вплоть до результатов валидации, к аналитическим данным, планам и отчетам применяли цифровые отметки времени в реальном времени.
[00312] План валидационного исследования
[00313] В первичном анализе случаи sPTB определяли как субъекты с родами из-за преждевременного разрыва околоплодных оболочек (PPROM) или самопроизвольного начала родоразрешения <37 0/7 недель GA. Контроли представляли собой субъектов, которые рожали на сроке≥37 0/7 недель GA. Перед анализами исследования и верификации исследовали 44 кандидатных биологических маркера, используя образцы сыворотки, собранные в широком интервале гестационного возраста (от 17 0/7 до 25 6/7 недель GA) (дополнительный материал). В исследовании и верификации идентифицировали оптимальный узкий интервал GA при взятии крови (от 19 0/7 до 21 6/7 недель) и два белка, IBP4 и SHBG, которые использовали в соотношении (IBP4/SHBG) в качестве наилучшего предиктора по AUROC для sPTB (дополнительный материал). В исследовании и верификации субъекты без экстремальных значений BMI улучшали эффективность классификации по IBP4/SHBG (дополнительные результаты). После анализов исследования и верификации авторы изобретения перешли к аналитической и клинической валидации.
[00314] В валидации случаев sPTB использовали всего 18 субъектов, собранных между 19 0/7 и 21 6/7 неделями GA при взятии крови (GABD), среди доступных всего 81 субъекта от 17 0/7 до 28 6/7 недель GA. Наборы контролей, содержащих два контроля на случай sPTB, которые совпадают по GABD, случайно отбирали, используя программу R Statistical (R 3.0.2) (Team RC. R: a Language and Environment for Statistical Computing. Vienna, Austria; 2014. 2015; Matei A, Tillé Y. The R «sampling» package. European Conference on Quality in Survey Statistics. 2006), и сравнивали с распределением полносрочных родов, как описано в 2012 National Vital Statistics Report (Martin JA, Hamilton BE, Osterman MJ, Curtin SC, Mathews TJ. Births: Final Data for 2012. National Vital Statistics Reports. 2014;63(09):1-86), используя критерий хи-квадрат. Случайно созданные контрольные наборы (в группах по 10) исследовали на наборы, дающие p-значение, приближающееся к 1,0.
[00315] Основная цель состояла в том, чтобы валидировать эффективность соотношения IBP4/SHBG в качестве предиктора для sPTB с использованием AUROC (Team RC. R: a Language and Environment for Statistical Computing. Vienna, Austria; 2014. 2015; Sing T, Sander O, Beerenwinkel N, Lengauer T. ROCR: visualizing classifier performance in R. Bioinformatics. 2005;21(20):7881). Для того чтобы контролировать общую частоту ошибок множественного тестирования (α=0,05), подход фиксированной последовательности (Dmitrienko A, Tamhane AC, Bretz F, ред. Multiple Testing Problems in Pharmaceutical Statistics. Boca Raton, Florida: CRC Press; 2009:1-320; Dmitrienko A, D'Agostino RB, Huque MF. Key multiplicity issues in clinical drug development. Stat Med. 2012;32(7):1079-111. doi:10.1002/sim.5642) применяли к GABD приращениям в оптимальном интервале (от 19 0/7 до 21 6/7 недель GA), идентифированном при исследовании и верификации, с применением BMI-стратификации и без нее (см. дополнительный материал). Значимость оценивали с помощью критерия Уилкоксона-Манна-Уитни, который тестирует эквивалентность относительно AUROC=0,5 (случайная вероятность). (Bamber D. The area above the ordinal dominance graph and the area below the receiver operating characteristic graph. Journal of mathematical psychology. 1975;12(4):387-415. doi:10.1016/0022-2496(75)90001-2; Mason SJ, Graham NE. Areas beneath the relative operating characteristics (ROC) and relative operating levels (ROL) curves: Statistical significance and interpretation. QJR Meteorol Soc. 2002;128(584):2145-2166. doi:10.1256/003590002320603584). Для определения эффективности классификации в границах GA, отличных от <37 0/7 в сравнении с≥37 0/7 недель GA (например, <36 0/7 в сравнении с≥36 0/7, <35 0/7 в сравнении с≥35 0/7), случаи и контроли переопределяли в качестве всех субъектов, которые ниже и равны/выше конкретной границы, соответственно.
[00316] Лабораторные способы
[00317] Подход системной биологии использовали для того, чтобы создавать высоко мультиплексный MS анализ мониторинга нескольких реакций (MRM) (дополнительные способы и результаты). Валидационный анализ количественно определял протеотипические пептиды, специфичные для предикторных белков IBP4 и SHBG и других контролей. Образцы обрабатывали партиями по 32, которые содержали клинические субъекты (24), объединенные сывороточные стандарты от здоровых не беременных доноров (HGS) (3), объединенные сывороточные стандарты от здоровых беременных доноров (pHGS) (3) и фосфатно-солевой буфер, который служил в качестве технологических контролей (2). Для всех анализов образцы сыворотки сначала истощали по не диагностическим белкам и белкам с высоким относительным содержанием с использованием иммунных колонок для истощения MARS-14 (Agilent Technologies), восстанавливали дитиотреитолом, алкилировали йодацетамидом и расщепляли трипсином. После этого сильно меченные стабильными изотопами стандартные (SIS) пептиды добавляли в образцы, которые впоследствии обессоливали и анализировали посредством жидкостной хроматографии с обращенной фазой (LC)/MRM-MS. SIS пептиды использовали для нормализации посредством создания коэффициентов ответа (RR), где площадь пика пептидного фрагментарного иона (т. е. переход), измеренную в сыворотке, делили на таковую для соответствующего SIS перехода, внесенного в тот же образец сыворотки.
[00318] Предиктор IBP4/SHBG
[00319] Оценку предиктора определяли как натуральный логарифм соотношения коэффициентов ответа переходов пептидов IBP4 и SHBG:
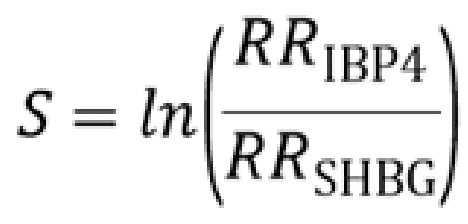
где RR представляют собой измеряемые коэффициенты ответа соответствующих пептидов.
[00320] РЕЗУЛЬТАТЫ
[00321] На фиг. 23 сведено распределение исследуемых субъектов в PAPR. Между мартом 2011 года и августом 2013 года в исследование введен 5501 субъект. Как предварительно определено в протоколе, 410 (6,7%) субъектов исключали из анализа из-за получения прогестогеновой терапии после первого триместра беременности. Дополнительные 120 (2,2%) субъектов исключены из-за раннего прерывания и для 146 (2,7%) утрачено наблюдение. Всего 4825 субъектов доступны для анализа. Сради них 533 PTB; 248 (4,7%) самопроизвольных и 285 (5,9%) с медицинскими показаниями. По сравнению с теми, кто рожал в срок, субъекты с sPTB с большей вероятностью имели одни или несколько предшествующих PTB и переносили кровотечение после 12 недель беременности в исследуемой беременности (таблица 1). Характеристики случаев sPTB и полносрочных контролей, отобранных для валидации, не значительно отличались друг от друга, за тем исключением, что имело место значительно больше испанских контролей (47,5% против 33,3% p=0,035). Аналогичным образом, субъекты, отобранные для валидации, главным образом представляли исследуемую когорту как целое (таблица 1), за исключением этнической принадлежности полносрочных контролей.
[00322] Валидационный анализ
[00323] В анализах исследования и верификации соотношение IBP4/SHBG и интервал между 19 0/7 и 21 6/7 неделями GA идентифицировали как наиболее эффективный предиктор sPTB по AUROC и GA интервалу, соответственно (дополнительные результаты, далее). Для валидации предварительно определенный подход фиксированной последовательности валидировал предиктор IBP4/SHBG с BMI-стратификацикй или без нее, с оптимальной эффективностью, идентифицированной для GA интервала от 19 1/7 до 20 6/7 недель. Не учитывая BMI, валидированная эффективность составляла AUROC=0,67 (p=0,02) (дополнительные результаты). Однако, как ожидалось, эффективность улучшали, используя BMI-стратификацию >22 и≤37 кг/м2, что соответствовало AUROC 0,75 (p=0,016, 95% CI 0,56-0,91) (фиг. 24). Более подробное определение характеристик BMI-стратификации можно найти в дополнительных результатах. Меры эффективности для чувствительности, специфичности, AUROC и отношений шансов (OR) определяли в варьирующих границах для случая в сравнении с контролем (таблица 2). Для sPTB в сравнении с полносрочными родами (<37 0/7 в сравнении с≥37 0/7 недель) чувствительность и специфичность составляли 0,75 и 0,74, соответственно, при отношении шансов (OR) 5,04 (95% CI 1,4-18). Результаты в других границах сведены в таблице 2. Точность теста улучшалась при более низких границах GA.
[00324] Скорректированное по болезненности положительное прогностическое значение (PPV), мера клинического риска, представлено как функция оценки предиктора на фиг. 25. Стратификация субъектов с возрастающей оценкой предиктора появляется с увеличением PPV от фонового значения (частота популяции sPTB 7,3% для одноплодных родов в США)( Martin et al., Births: final data for 2013. Natl Vital Stat Rep. 2015;64(1):1-65 Martin JA, Hamilton BE, Osterman MJ, Curtin SC, Matthews TJ. Births: final data for 2013. Natl Vital Stat Rep. 2015;64(1):1-65) до относительных рисков 2× (14,6%) и 3× (21,9%) (штриховые линии) и выше (фиг. 25). Распределение значений оценки предиктора IBP4/SHBG для субъектов с цветовым кодированием категории по GA при родах представлено в диаграммах размаха на фиг. 25. Самые ранние случаи sPTB (<35 0/7 недель GA) имеют более высокие оценки предиктора, чем поздние полносрочные контроли (≥39 0/7 недель GA), хотя оценки для поздних случаев sPTB (от≥35 0/7 до <37 0/7 недель GA) перекрываются с ранними полносрочными контролями (от≥37 0/7 до <39 0/7 недель GA) (фиг. 25). Валидационных субъектов идентифицировали как высокий или низкий риск в соответствии с порогом оценки предиктора, соответствующим 2× относительному риску (PPV 14,6%). Затем частоту родов для групп высокого и низкого риска отображали как события в анализе Каплана-Мейера (фиг. 26). Из этого анализа, те, которых классифицировали как высокий риск, в целом рожали раньше, чем те, которых классифицировали как низкий риск (p=0,0004).
[00325] Поствалидационный анализ
[00326] Эффективность предиктора измеряли с использованием комбинации субъектов из слепой верификации (дополнительные данные, далее) и валидационных анализов в оптимальном интервале BMI и GA. ROC-кривая для комбинированного набора образцов представлена и соответствует AUROC 0,72 (p=0,013) (фиг. 27).
[00327] Используя «-омный» подход, авторы изобретения разработали предиктор материнской сыворотки, состоящий из соотношения уровней IBP4/SHBG в 19-20 недели с интервалом BMI >22 и≤37 кг/м2, который идентифицировал 75% женщин, у которых будут sPTB. Предшествующий анамнез sPTB (Goldenberg et al., Epidemiology and causes of preterm birth. Lancet. 2008;371(9606):75-84. doi:10.1016/S0140-6736(08)60074-4, Petrini et al. Estimated effect of 17 alpha-hydroxyprogesterone caproate on preterm birth in the United States. Obstet Gynecol. 2005;105(2):267-272) и измерения длины шейки матки (Iams et al. The length of the cervix and the risk of spontaneous premature delivery. National Institute of Child Health and Human Development Maternal Fetal Medicine Unit Network. N Engl J Med. 1996;334(9):567-72; Hassan et al. Vaginal progesterone reduces the rate of preterm birth in women with a sonographic short cervix: a multicenter, randomized, double-blind, placebo-controlled trial. Ultrasound Obstet Gynecol. 2011;38(1):18-31) считают наилучшими мерами клинического риска на сегодняшний день; однако индивидуально или в комбинации они не способны предсказать большинство sPTB.
[00328] Идеальный инструмент предсказания sPTB должен быть минимально инвазивным, работать при ранней беременности, совпадая по времени с обычными акушерскими приемами, и точно идентифицировать тех, кто имеет самый высокий риск. Существующие «-омные» исследования показывают, что нарушения в физиологическом состоянии беременности можно обнаруживать в анализируемых веществах материнской сыворотки, измеряемых у sPTB субъектов. «-омные» исследования при PTB включали протеомные (Gravett et al. Proteomic analysis of cervical-vaginal fluid: identification of novel biomarkers for detection of intra-amniotic infection. J Proteome Res. 2007;6(1):89-96; Goldenberg et al. The preterm prediction study: the value of new vs standard risk factors in predicting early and all spontaneous preterm births. NICHD MFMU Network. Am J Public Health. 1998;88(2):233-8; Gravett et al. Diagnosis of intra-amniotic infection by proteomic profiling and identification of novel biomarkers. JAMA. 2004;292(4):462-469; Pereira et al. Insights into the multifactorial nature of preterm birth: proteomic profiling of the maternal serum glycoproteome and maternal serum peptidome among women in preterm labor. Am J Obstet Gynecol. 2010;202(6):555.e1-10; 32. Pereira et al. Identification of novel protein biomarkers of preterm birth in human cervical-vaginal fluid. J Proteome Res. 2007;6(4):1269-76; Dasari et al. Comprehensive proteomic analysis of human cervical-vaginal fluid. J Proteome Res. 2007;6(4):1258-1268; Esplin et al. Proteomic identification of serum peptides predicting subsequent spontaneous preterm birth. Am J Obstet Gynecol. 2010;204(5):391.e1-8), транскриптомные (Weiner et al. Human effector/initiator gene sets that regulate myometrial contractility during term and preterm labor. Am J Obstet Gynecol. 2010;202(5):474.e1-20; Chim et al. Systematic identification of spontaneous preterm birth-associated RNA transcripts in maternal plasma. PLoS ONE. 2012;7(4):e34328. Enquobahrie et al. Early pregnancy peripheral blood gene expression and risk of preterm delivery: a nested case control study. BMC Pregnancy Childbirth. 2009;9(1):56), геномные (Bezold et al. The genomics of preterm birth: from animal models to human studies. Genome Med. 2013;5(4):34; Romero et al. Identification of fetal and maternal single nucleotide polymorphisms in candidate genes that predispose to spontaneous preterm labor with intact membranes. Am J Obstet Gynecol. 2010;202(5):431.e1-34; Swaggart et al. Genomics of preterm birth. Cold Spring Harb Perspect Med. 2015;5(2):a023127; Haataja et al. Mapping a new spontaneous preterm birth susceptibility gene, IGF1R, using linkage, haplotype sharing, and association analysis. PLoS Genet. 2011;7(2):e1001293; McElroy et al. Maternal coding variants in complement receptor 1 and spontaneous idiopathic preterm birth. Hum Genet. 2013;132(8):935-42) и метаболомные (Menon et al. Amniotic fluid metabolomic analysis in spontaneous preterm birth. Reprod Sci. 2014;21(6):791-803) подходы. Однако для датирования ни один из этих подходов не дал валидированных способов тестирования для надежного предсказания риска sPTB у бессимптомных женщин.
[00329] Данное изобретение являвется результатом большого проспективного и современного клинического исследования, которое сделало возможным независимый исследовательский, верификационный и валидационный анализ, при этом придерживаясь руководств IOM, касающихся разработки «-омных» тестов. Он включает построение большого и стандартизованного мультиплексного протеомного анализа для исследования биологических путей, имеющих отношение к беременности. Масштаб исследования и относительно широкий интервал взятия крови (от 17 0/7 до 28 6/7 недель GA) также сделали возможной идентификацию GA интервала, в котором отмечены изменения концентраций белков между случаями sPTB и полносрочными контролями. Использование модели предиктора низкой сложности (т. е. соотношения двух белков) ограничило опасности чрезмерной подгонки.
[00330] Применение протеомного анализа и построение модели привело к идентификации пары критичных белков (IBP4 и SHBG) с уверенно хорошей предсказательной эффективностью для sPTB. Несмотря на сложности при построении классификатора для состояния, которому приписывают множество этиологий, предиктор демонстрировал хорошую эффективность при пороге <37 0/7 в сравнении с≥37 0/7 недель GA с AUROC 0,75. Важно, что точность предиктора повышается для более ранних sPTB (например <35 0/7 недель GA), что делает возможным обнаружение этих sPTB с наибольшим потенциалом для распространенности болезни. Субъекты, у которых определяли высокий риск sPTB, используя предиктор IBP4/SHBG, рожали значительно раньше, чем субъекты, у которых идентифицировали низкий риск. Находки авторов изобретения подсказывают, что IBP4 и SHBG могут выполнять важные функции, связанные с этиологиями sPTB, и/или действовать в качестве точек сходимости в релевантных биологических путях.
[00331] Универсальное трансвагинальное ультразвуковое (TVU) измерение длины шейки матки (CL) осуществляли обычным образом в большинстве исследовательских центров, использованных в изобретении, и оно было доступно для меньше чем 1/3 исследуемых субъектов. Будет интересно оценивать, усовершенствуют ли измерения CL протеомный предиктор в будущих исследованиях, или, альтернативно, рисковая стратификация с помощью классификатора IBP4/SHBG идентифицирует женщин, которые больше всего выиграют от серийных измерений CL. Наконец, будет увлекательно исследовать эффективность молекулярного предиктора вместе с переменной BMI или, возможно, в комбинации с другими характеристиками медицинского анамнеза/анамнеза беременности и социальной демографии.
[00332] В заключение, предварительно определяемый предсказательный тест для sPTB на основании измерений IBP4 и SHBG в сыворотке у бессимптомных рожавших и нерожавших женщин валидировали в полностью независимом наборе субъектов. Дополнительные функциональные исследования этих белков, регуляции их генов и связанных путей могут помочь прояснить молекулярные и физиологические основы sPTB. Применение этого предиктора должно сделать возможным раннее и чувствительное обнаружение женщин с риском sPTB. Это может улучшать исходы беременности через усиленное клиническое наблюдение, а также ускорять разработку клинического вмешательства для предотвращения PTB.
[00333] ДОПОЛНИТЕЛЬНЫЕ МАТЕРИАЛЫ И СПОСОБЫ
[00334] Субъекты исследования и верификации
[00335] Субъектов исследования и верификации брали из PAPR исследования, описанного выше в этом примере.
[00336] Принципы исследования и верификации
[00337] В этом примере определяли случаи sPTB, как описано выше. Исследование и верификацию предиктора проводили в соответствии с руководством по передовому опыту в «-омных» исследованиях. (IOM (Institute of Medicine). Evolution of Translation Omics: Lessons Learned and the Path Forward. (Micheel CM, Nass SJ, Omenn GS, ред.). Washington, DC: The National Academies Press.; 2012:1-355). В кмбинированном анализе случаев/контролей использовали наборы образцов, полностью независимые друг от друга. Случаи и контроли, отобранные для исследования и верификации, проходили централизованное рассмотрение на предмет разногласий в данных субъекта; не осуществляли верификацию исходных документов (SDV) с медицинской картой. Все случаи sPTB и контроли для исследования и верификации индивидуально корректировал главный медицинский специалист и разногласия проясняли с помощью PI в клинической локализации. Детализирвоанные протоколы анализа, включая планы исследований, планы анализа и протоколы слепой верификации были созданы предварительно. Персонал лабораторий и анализа данных вслепую верифицировал присвоение данных о случае субъекта, контроле и GA. Оценки предиктора, вычисленные, как описано ниже, присваивали всем субъектам посредством внутренней слепой статистики. Данные о случае, контроле и GA, связанные с оценками предиктора с помощью DCO, предоставляли независимому внешнему статистику для анализа. Затем результаты AUROC передавали обратно к DCO. При передаче данных использовали функцию SUMPRODUCT (Microsoft. Microsoft Excel. 2013) в Excel, чтобы гарантировать сохранение целостности данных. Чтобы предоставлять контрольный журнал данных от каждого субъекта вплоть до результатов валидации, к аналитическим данным, планам и отчетам применяли цифровые отметки времени в реальном времени.
[00338] План исследования и верификации
[00339] В исследовании и верификации случаев sPTB собирали всего 86 и 50 субъектов, соответственно, от 17 0/7 до 28 6/7 недель GA при взятии крови (GABD). Субъекты, которых использовали в исследовании и верификации, были полностью независимыми друг от друга и независимыми от тех, кого использовали в валидации. Идентифицировали совпадающие контроли для случаев sPTB в исследовании и верификации, как описано выше в этом примере.
[00340] Анализ болезненности
[00341] После исследовательского, верификационного и валидационного анализа, дополнительные полносрочные контроли, не использованные в предыдущих исследованиях, выбирали из базы данных PAPR и обрабатывали в лаборатории с использованием MRM-MS анализа, который применяли при валидации и который описан выше в этом примере. Используя пакет Sampling в программном обеспечении R Statistical (версия 3.0.3) (Team RC. R: a Language and Environment for Statistical Computing. Vienna, Austria; 2014. 2015; Matei A, Tillé Y. The R «sampling» package. European Conference on Quality in Survey Statistics. 2006), наборы из 187 субъектов случайно выбирали из валидированного GA интервала взятия крови и сравнивали через одномерный статистический анализ (критерий хи-квадрат) в сравнении с данными о гестационном возрасте при рождении (GAB) из 2012 National Vital Statistics Report (NVSR). Martin et al.: Final Data for 2012. National Vital Statistics Reports. 2014;63(09):1-86 Затем отбирали наборы контролей, наиболее близко приближенные к распределению родов в 2012 NVSR на основании наилучшего p-значения (стремящегося к 1,0 с минимальным приемлемым значением 0,950) для сравнения с BMI распределением в исследовании PAPR в целом. Используя одномерный статистический анализ (критерий хи-квадрат) в сравнении с BMI данными из базы данных исследования PAPR, наборы контролей, наиболее близко приближенные к распределению BMI (стремящиеся к 1,0 с минимальным приемлемым значением 0,950) и распределению времени родов в NVSR, отбирали и сравнивали с GABD валидированных образцов взятия крови. Набор, наиболее близко приближенный ко всем трем распределениям, выбирали в качестве набора субъектов для исследования болезненности. Значения оценки предиктора для верификации, валидации и болезненности в пределах валидированного интервала GABD и BMI ограничения объединяли 150 субъектов. Этот составной массив данных использовали для получения наилучших оценок доверительных областей около кривой PPV на фиг. 25. Доверительные области около PPV вычисляли с использованием нормальной аппроксимации ошибки для биномиальных долей. Brown et al. Interval estimation for a binomial proportion. Statistical science. 2001;16(2):101-133.
[00342] Лабораторные способы
[00343] Подход системной биологии использовали для того, чтобы создавать высоко мультиплексный масс-спектрометрический (MS) анализ мониторинга нескольких реакций (MRM) посредством итерационного применения: литературного курирования, нацеленного и не нацеленного протеомного исследования и мелкомасштабного MRM-MS анализа образцов субъектов. Полный MRM-MS анализ с измерением 147 белков применяли в исследовании и верификации. Для всех анализов, образцы сыворотки обрабатывали в лаборатории, как описано выше в этом примере. Аликвоты объединенных сывороточных контролей (pHGS) использовали для того, чтобы вычислять аналитический коэффициент вариации (CV) между партиями для IBP4 и SHBG.
[00344] Основная стратегия разработки предиктора
[00345] Разрабатывали стратегию для того, чтобы избегать чрезмерной подгонки и преодолевать ослабление эффективности биологического маркера, ожидаемых в широких диапазонах гестационного возраста из-за динамической природы экспрессии белков во время беременности. При разработке предиктора использовали соотношения интенсивностей анализируемых веществ под повышающей регуляцией и под понижающей регуляцией. Такие «обращения» схожи со стратегиями с парами с наивысшей оценкой и 2-генными классификаторами. (Geman et al. Classifying gene expression profiles from pair wise mRNA comparisons. Stat Appl Genet Mol Biol. 2004;3(1):Article19; Price et al. Highly accurate two-gene classifier for differentiating gastrointestinal stromal tumors and leiomyosarcomas. Proc Natl Acad Sci USA. 2007;104(9):3414-9). Этот подход делает возможным усиление диагностического сигнала и самостоятельную нормализацию, поскольку оба белка в «обращении» проходят те же стадии преданалитической и аналитической обработки. В качестве стратегии для нормализации мер интенсивности пептидов на сложных протеомных технологических маршрутах, обращения также схожи со введенным в последнее время подходом, называемым «нормализация эндогенных белков (EPN)». (Li et al. An integrated quantification method to increase the precision, robustness, and resolution of protein measurement in human plasma samples. Clin Proteomics. 2015;12(1):3; Li et al. A blood-based proteomic classifier for the molecular characterization of pulmonary nodules. Sci Transl Med. 2013;5(207):207ra142). Число кандидатных анализируемых веществ, используемых для построения модели, снижали с помощью аналитических критериев. Аналитические фильтры включали: пороги для аналитической воспроизводимости, интенсивности, свидетельства о взаимодействии, зависимости от порядка обработки образцов и преданалитической стабильности. Общее число анализируемых веществ в каком-либо одном предикторе было ограничено одним обращением, чтобы, таким образом, избегать сложных математических моделей. Оценки предиктора определяли как натуральный логарифм одного значения обращения, где само обращение представляет собой коэффициент ответа (который определен выше в этом примере). Наконец, предсказательную эффективность изучали в узких перекрывающихся 3-недельных интервалах беременности.
[00346] Кривые рабочей характеристики приемника
[00347] AUROC значения и связанные p-значения вычисляли для обращений, как описано выше в этом примере. Распределение и среднее значение для предиктора AUROC в комбинированном наборе для исследования и верификации вычисляли с использованием бутстрэп-выборки, выполняемой итерационно посредством выбора случайных наборов образцов с заменой. Efron B, Tibshirani RJ. An Introduction to the Bootstrap. Boca Raton, Florida: Chapman and Hall/CRC Press; 1994. Общее число отобранных образцов в каждой итерации соответствовало общему доступному в начальной совокупности.
[00348] ДОПОЛНИТЕЛЬНЫЕ РЕЗУЛЬТАТЫ
[00349] Исследовательские, верификационные и валидационные характеристики субъекта сведены в таблице 3. Процентная доля субъектов с одними или несколькими предшествующими sPTB в исследовательских случаях sPTB была выше, чем при верификации или валидации, и другие характеристики, главным образом, были согласованными между исследованиями.
[00350] Исследовательский и верификационныйц анализ
[00351] Сорок четыре белка находились под повышающей или понижающей регуляцией в перекрывающихся 3-недельных GA интервалах и проходили аналитические фильтры (фиг. 28). Обращения формировали из соотношениий белков под повышающей и понижающей регуляцией и тестировли предсказательную эффективность в образцах в каждом из перекрывающихся 3-недельных GA интервалов. Эффективность для поднабора обращений, отражающая репрезентативные паттерны, представлена на фиг. 29. Волны эффективности очевидны: обращения IBP4/SHBG и APOH/SHBG обладали более хорошими значениями AUROC в ранних интервалах, тогда как ITIH4/BGH3 и PSG2/BGH3 достигали пика позже в беременности (фиг. 24). Некоторые обращения имели согласованную, но умеренную эффективность по всему диапазону гестационного возраста (PSG2/PRG2) (фиг. 29). Наиболее эффективное обращение из всех, IBP4/SHBG, имело AUROC=0,74 в интервале от 19 0/7 до 21 6/7 (фиг. 29). AUROC эффективность предиктора IBP4/SHBG возрастала до 0,79, когда субъектов стратифицировали по BMI до беременности <35 (кг/м2) (таблица 4). В силу его согласованно высокой эффективности при ранней беременности (т. е. от 17 0/7 до 22 6/7 недель GA) (фиг. 29) и потенциально желательной клинической полезности, предиктор IBP4/SHBG выбирали для верификационного анализа.
[00352] Слепая IBP4/SHBG AUROC эффективность на верификационных образцах составляла 0,77 и 0,79 для всех субъектов и BMI-стратифицированных субъектов, соответственно, в хорошем соответствии с эффективностью, получаемой в исследовании (таблица 5). После слепой верификации, исследовательские и верификационные образцы комбинировали для определения эффективности бутстрэпа. Среднюю AUROC 0,76 получали после 2000 итераций бутстрэпа (фиг. 30).
[00353] BMI валидационный анализ
[00354] Эффективность предиктора IBP4/SHBG оценивали при нескольких порогах BMI в валидационных образцах (таблица 5). AUROC измеренная эффективность немного улучшалась посредством элиминации или очень высокого (например >37 кг/м2) или низкого BMI (например≤22 кг/м2). Стратификация посредством комбинации этих двух порогов давала AUROC 0,75 (таблица 5).
Пример 3. Корреляция данных масс-спектрометрического и иммунологического анализа
[00355] Этот пример демонстрирует результаты скрининга Myriad RBM, идентифицирующего IBP4 и другие биологические маркеры индивидуальные биологические маркеры для sPTB при раннем, среднем и позднем интервалах гестационного возраста при взятии, (2) корреляцию результатов MS и иммунологического анализа для SHBG/IBP4 и (3) клинические данные, связанные с SHBG, в качестве биологического маркера для sPTB.
[00356] Данные RBM
[00357] В кратком изложении, в RBM анализировали 40 случаев и 40 контролей из PAPR (20/20 из раннего интервала), 10/10 из среднего интервала, 10/10 из позднего интервала). В RBM использовали Human Discovery MAP 250+ v2.0 (Myriad RBM, Austin, TX). Цель этого анализа состоит в том, чтобы разработать многомерные модели для того, чтобы предсказать PTB, используя множество анализируемых веществ. Авторы изобретения использовали четыре способа моделирования: случайный лес (rf), бустинг, лассо и логистический (logit). Авторы изобретения выполняли первый раунд отбора переменных, в котором каждым способом независимо выбирают 15 наилучших переменных для этого способа. Из 15 отбирали наилучшие анализируемые вещества независимо с помощью каждого из четырех способов моделирования, используя пошаговое исключение и оценку площади под ROC-кривой (AUC) с использованием исключенных из бутстрэпа образцов. В таблице 6 представлены наилучшие результаты из нескольких многопараметрических моделей. В таблице 7 представлено ранжирование анализируемых веществ в раннем интервале (GABD 17-22 нед.) с помощью различных многомерных моделей. В таблице 8 представлено ранжирование анализируемых веществ в среднем интервале (GABD 23-25 нед.) с помощью различных многомерных моделей. В таблице 9 представлено ранжирование анализируемых веществ в позднем интервале (GABD 26-28 недели) с помощью различных многомерных моделей.
[00358] Идентификация коммерческих комплектов ELISA, которые коррелируют с масс-спектрометрическими данными
[00359] В кратком изложении, сравнение ELISA с MS включало множество исследований с использованием PAPR образцов и находилось в диапоне размеров 30-40 субъектов. Каждый ELISA осуществляли в соответствии с протоколом изготовителя. Затем предсказанную с помощью ELISA концентрацию каждого анализируемого вещества сравнивали с полученными MS удельными соотношениями от идентичных образцов. Затем для сравнения генерировали значение r корреляции Пирсона. Сравнение ELISA с MS включало множество исследований с использованием PAPR образцов и находилось в диапазоне размеров 30-40 субъектов. Каждый ELISA осуществляли в соответствии с протоколом изготовителя. Затем предсказанную с помощью ELISA концентрацию каждого анализируемого вещества сравнивали с полученными MS удельными соотношениями от идентичных образцов. Затем для сравнения генерировали значение r корреляции Пирсона. В таблице 10 представлена информация об эпитопах и клональности для комплектов, тестированных по анализируемым веществам IBP4_HUMAN И SHBG_HUMAN. В таблице 11 показано, что не все наборы для ELISA коррелируют с MS, даже для белков, где корреляция существует. См., например: IBP4, CHL1, ANGT, PAPP1.
[00360] 120 предварительно замороженных образцов сыворотки с известными исходами из исследования PAPR отбирали для сравнения между анализами ELISA и MS. Эти образцы имеют гестационный возраст при взятии крови (GABD) между 119 и 180 сутками. Образцы не исключали из-за материнского BMI. ELISA осуществляли на коммерчески доступных комплектах для IBP4 (AL-126, ANSCH Labs Webster, Texas) и SHBG (DSHBG0B, R&D Systems Minneapolis, Minnesota). Анализы проводили в соответствии с протоколами производителей. Внутренние стандарты использовали для нормализации от планшета к планшету. Оценку вычисляли по ELISA значениям концентрации в соответствии с LN([IBP4]/[SHBG]), и по MS в соответствии с LN(IBP4RR/SHBGRR), где RR относится к удельному соотношению площадей пиков эндогенного пептида и SIS пептида. Оценки, полученные из двух подходов сравнивали при разделении случая и контролея (p-значения, полученные из критерия Стьюдента для одной выборки, принимая равные стандартные отклонения) (фиг. 31).
[00361] Пятьдесят семь предварительно замороженных образцов сыворотки (19 случаев sPTB, 38 полносрочных контролей) с известными исходами из исследования PAPR отбирали для сравнения между анализами ELISA и MS. Эти образцы имеют гестационный возраст при взятии крови (GABD) между 133 и 148 сутками. ELISA осуществляли на коммерчески доступных комплектах для IBP4 (AL-126, ANSCH Labs Webster, Texas) и SHBG (DSHBG0B, R&D Systems Minneapolis, Minnesota). Анализы проводили в соответствии с протоколами производителей. Образцы, использованные в различных планшетах, нормализовали с использованием внутренних стандартов. Оценку вычисляли по ELISA значениям концентрации в соответствии с LN([IBP4]/[SHBG]) и по MS в соответствии с LN(IBP4RR/SHBGRR), где RR относится к удельному соотношению площадей пиков эндогенного пептида и SIS пептида. Затем определяли эффективность иммунологического анализа с помощью площади под кривой рабочей характеристики приемника (AUC) и сравнивали с полученной MS AUC на тех же наборах образцов (фиг. 32). Значения AUC также определяли после применения BMI-стратификации к образцам (BMI >22≤37), что давало всего 34 образца (13 случаев sPTB, 21 полносрочный контроль) (фиг. 33).
[00362] 60 предварительно замороженных образцов сыворотки с известными исходами из исследования PAPR анализировали посредством анализов ELISA и MS. Эти образцы имели предсказанный гестационный возраст при взятии крови (GABD) между 133 и 146 сутками. Корреляционный анализ осуществляли для образцов при всех BMI (фиг. 34, правая часть) или для поднабора образцов с BMI >22 или≤37 (фиг. 34, левая часть). ELISA осуществляли на коммерчески доступных комплектах для IBP4 (AL-126, ANSCH Labs Webster, Texas) и SHBG (DSHBG0B, R&D Systems Minneapolis, Minnesota). Анализ проводили в соответствии с протоколами производителей. Внутренние стандарты использовали для нормализации от планшета к планшету. Оценку вычисляли по ELISA значениям концентрации в соответствии с LN([IBP4]/[SHBG]) и по MS в соответствии с LN(IBP4RR/SHBGRR), где RR относится к удельному соотношению площадей пиков эндогенного пептида и SIS пептида. Оценки, полученные из двух подходов, сравнивали посредством корреляции и при разделении случая и контроля (p-значения, полученные из критерия Стьюдента для одной выборки, предполагая равные стандартные отклонения). В таблице 12 представлены комплекты ELISA для IBP4 и SHBG, демонстрирующие разделение (одномерное) sPTB и контроля.
[00363] Сравнение измерений SHBG с помощью масс-спектрометрии и клинических анализаторов
[00364] 35 образцов от индивидуальных субъектов и объединенные сыворотки беременных и не беременных женщин одновременно анализировали в Sera Prognostics и двух независимых справочных лабораториях, ARUP Laboratories и Intermountain Laboratory Services. Аликвоты транспортировали в каждую лабораторию охлажденными и доставку координировали так, что тесты начинали в одну и ту же дату для всех трех лабораторий. В ARUP использовали анализатор Roche cobas e602*, а в Intermountain использовали Abbott Architect CMIA, оба являются полуавтоматизированными приборами иммунологического анализа. В Sera Prognostics использовали уникальный способ протеомного анализа, включающий иммунное истощение образцов, ферментативное расщепление и анализ на масс-спектрометре Agilent 6490. Результаты из ARUP и IHC приведены в нмоль/л, тогда как в Sera использовали удельное соотношение (RR) тяжелых и легких пептидных суррогатов. Данные из ARUP и Intermountain сравнивали друг с другом для того, чтобы определять точность (фиг. 39). Линейность и воспроизводимость хорошо соответствовали на всем протяжении широкого диапазона результатов с наклоном линейности 1,032 и значением r2 0,990. Затем данные из каждой справочной лаборатории сравнивали с RR из Sera и графиком линейной регрессии (фиг. 37 и 38). Данные хорошо совпадали с результатами Sera с ARUP, имеющей значение r2 0,937, и Intermountain, имеющей значение r2 0,934.
Пример 4. SNP, инсерции и делеции и структурные варианты в пептидах PreTRM IBP4 и SHBG
[00365] В этом примере показаны известные SNP, инсерции и делеции (инсерции-делеции) и структурные варианты в пептидах PreTRM IBP4 и SHBG.
[00366] В таблице 13 и таблице 14 детализированы известные SNP, инсерции и делеции (инсерции-делеции) и структурные варианты в пептидах PreTRM IBP4 и SHBG. Информацию извлекают из базы данных об однонуклеотидных полиморфизмах (dbSNP), сборка 146. Одна миссенс вариация (G>C) в SHBG, A179P (dbSNP id: rs115336700) имеет наибольшую общую аллельную частоту 0,0048. Хотя эта аллельная частота низка, несколько субпопуляций, изученных в проекте 1000 Genomes, имели значительно более высокие частоты. Этими популяциями (аллельные частоты) являются: американцы африканского происхождения в SW USA (0,0492); карибские африканцы в Барбадосе (0,0313); юрба в Ибадане, Нигерия (0,0278); лухья в Вебуйе, Кения (0,0101); есан в Нигерии (0,0101); колумбийцы из Медельина, Колумбия (0,0053); гамбийцы в западных регионах Гамбии (0,0044). Все другие изученные субпопуляции не имеют вариаций в этом нуклеотидном положении. Заголовок таблицы содержит id кластера - (номер dbSNP rs), гетерозиготность - усредненную гетерозиготность, валидацию - способ валидации (или пустой без валидации), MAF - частоту минорного аллеля, функцию - функциональную характеристику полиморфизма, аллель dbSNP - отличительные черты аллельного нуклеотида, остаток белка - остаток, являющийся результатом аллеля, кодон. полож. -положение в кодоне, аминокислотн. полож. в NP_001031.2 - аминокислотное положение в эталонной последовательности NP_001031.2 и мРНК полож. в NM_001040.2 - нуклеотидное положение в эталонной последовательности NM_001040.2.
Пример 5. Обращение IBP4/SHBG усиливает диагностический сигнал для sPTB и снижает аналитическую вариабельность
[00367] Этот пример демонстрирует усиление диагностического сигнала и снижение вариабельности, достигаемые с использованием стратегии обращения IBP4/SHBG.
[00368] Представлены уровни IBP4 и SHBG, которые определяли с помощью MS в указанном диапазоне сроков беременности для случаев sPTB и полносрочных контролей отдельно (фиг. 44 и фиг. 45). Кривые создавали посредством сглаживания средней удельных соотношений пептидов (площади пика эндогенного пептида и соответствующей SIS площади пика). Сигнал случая в сравнении с контролем соответствует приблизительной максимальной 10% разности для IBP4 и SHBG. При построении графика оценки, вычисленной как ln(IBP4RR/SHBGRR), усиление сигнала очевидно (максимальная разность приблизительно 20%) (фиг. 46). Эти данные демонстрируют усиление диагностического сигнала, получаемое с использованием стратегии обращения IBP4/SHBG.
[00369] Формирование соотношения уровней двух белков может снижать вариабельность, поскольку каждый белок проходит одни и те же стадии аналитической и преданалитической обработки. Чтобы исследовать влияние на вариабельность, определяли CV для индивидуальных белков (RR для IBP4 и SHBG) и для соотношения IBP4RR/SHBGRR в объединенных образцах сывороток контролей от беременных доноров (pHGS). Объединенные контрольные образцы, лишенные биологической вариабельности, анализировали в нескольких партиях и на протяжении нескольких суток. Вариабельность обращения меньше вариабельности, связанной с индивидуальными белками (фиг. 48).
[00370] Чтобы исследовать, усиливает ли формирование обращения в целом диагностический сигнал, авторы изобретения исследовали ROC эффективность (AUC) высоко эффективных обращений (AUC >0,6), сформированных посредством соотношения многих белков. В верхней части на фиг. 47 представлен диапазон значений AUC (случай sPTB в сравнении с полносрочным контролем) с использованием массивов данных от образцов, собранных между 19/0 неделями и 21/6 неделями беременности. Смежные диаграммы размаха показывают диапазон ROC эффективности для индивидуальных белков под повышающей регуляцией и понижающей регуляцией, используемых для того, чтобы формировать связанные обращения. Аналогичным образом, p-значения, полученные из критерия Уилкоксона (случай sPTB в сравнении с полносрочными контролями) для обращений, более значимы, чем таковые для соответствующих индивидуальных белков (фиг. 47, внизу).
[00371] Чтобы исследовать, снижает ли формирование обращений в более общем смысле вариабельность, авторы изобретения исследователи аналитическую вариабельность для 72 различных значений обращения (т. е. соотношение относительных площадей пиков в сравнении с аналитической вариабельностью индивидуальных белков, которые содержат обращения в объединенных образцах сывороток контролей от беременных доноров (pHGS). Объединенные контрольные образцы, лишенные биологической вариабельности, анализировали в нескольких партиях и на протяжении нескольких суток. Вариабельность обращения меньше вариабельности, связанной с индивидуальными белками (фиг. 49).
[00372] Генерализуемость стратегии обращения для того, чтобы снижать аналитическую вариабельность.
[00373] На фиг. 48 приведены CV, вычисленные для pHGS образцов (объединенных образцов беременных), которые анализировали в лаборатории несколькими партиями, в несколько суток и на нескольких приборах. Поскольку CV вычисляли с использованием pHGS образцов, которые лишены биологической вариабельности, они соответствуют мере аналитической вариабельности, вводимой при лабораторной обработке образцов. Аналитическая вариабельность, связанная со значением, использованном в соотношении, для 72 обращений, ниже аналитической вариабельности относительных площадей пиков индивидуальных белков под повышающей регуляцией и понижающей регуляцией, используемых для того, чтобы формировать обращения (фиг. 49).
Пример 6. Анализ медицински показанных PTB
[00374] Этот пример подтверждает, что классификатор чувствителен к компонентам медицински показанных PTB на основании состояний, таких как предэклампсия или диабет беременных.
[00375] PreTRMTM разрабатывали и валидировали в качестве предиктора для самопроизвольных PTB. Приблизительно 75% всех PTB в США являются самопроизвольными, остальные являются медицински показанными из-за нескоторых осложнений у матери или плода (например, предэклампсия, задержка внутриутробного развития, инфекция). 41 образец медицински показанных PTB из биобанка PAPR анализировали в лаборатории и вычисляли оценки PreTRM. Оценки PreTRMTM сравнивали для субъектов, аннотированных в качестве имеющих медицинские показания для предэклампсии в сравнении с другими показаниями. Субъекты с медицинскими показаниями к преждевременному родоразрешению по причине предэклампсии имели значительно более высокие оценки, чем другие (фиг. 50).
[00376] На фиг. 52 представлена тепловая карта интенсивности обращений с аннотацией для диабета. Красными стрелками показаны субъекты с диабетом. Образцы перечислены внизу, где случаи PTB находятся в правой части скрининга и полносрочные роды в левой части. Пациенты с диабетом кластеризованы справа, что показывает, что можно идентифицировать обращения, которые стратифицируют диабет беременных и, таким образом, которые позволяют создать диагностический тест по биологическим маркерам для предсказания диабета беременных.
Пример 7. Другие переходы и пептиды
[00377] В таблице 16 представлены сравнительные данные для пептидов и MS переходов IBP4. Четыре различных интенсивно меченных пептида (R*+10 Да) служат примером различных переходов и их относительных интенсивностей, мониторинг которых можно осуществлять для того, чтобы количественно определять IBP4. Специалисты в данной области могут выбирать потенциально любые из этих пептидов или переходов или других, не приведенных в качестве примера, чтобы количественно определять IBP4.
[00378] В таблице 17 представлены сравнительные MS данные для пептидов и переходов IBP4. Триптические пептиды IBP4, полученные из рекомбинантного белка, анализировали посредством MRM-MS для того, чтобы идентифицировать кандидатный суррогатный пептид и их переходы. Специалисты в данной области могут выбирать потенциально любые из этих пептидов или переходов или других, не приведенных в качестве примера, чтобы количественно определять IBP4. IBP4 идентифицировали в RBM (выше), затем заказывали синтетический пептид для создания анализа.
[00379] В таблице 18 представлены сравнительные данные для пептидов и MS переходов SHBG. Триптические пептиды SHBG, полученные из рекомбинантного белка или объединенной сыворотки беременных, анализировали посредством MRM-MS, чтобы идентифицировать кандидатный суррогатный пептид и их переходы. Специалисты в данной области могут выбирать потенциально любые из этих пептидов или переходов или других, не приведенных в качестве примера, чтобы количественно определять SHBG. Также показаны специфичные пептиды изоформ, которые идентифицировали в сыворотке.
[00380] В таблице 19 представлены белки с измененными уровнями в сыворотке на протяжении 17-25 недель GA в PTB образцах. * Дополнительные белки, ограниченные неделями 19-21 GA в PTB. LC-MS (MRM) анализ 148 белков для нескольких путей и анализированных образцов сыворотки из 17-25 недель гестационного возраста (GA) от 312 женщин (104 случая sPTB, 208 полносрочных контролей). Данные MRM площади пика анализировали с помощью иерархической кластеризации, критериев Стьюдента и зависимости от GA. После аналитической фильтрации 25 белков демонстрировали значимые различия (p<0,05) у субъектов с sPTB в сравнении с полным сроком (таблица 1). Уровни 14 белков были выше и 3 были ниже в sPTB образцах во всем диапазоне GA. Обнаружены другие белки, подлежащие динамической регуляции в частичных интервалах периода GA. Например, в недели GA 19-21 дополнительные 7 белков повышены и 1 понижен при sPTB.
[00381] В таблице 20 перечислены 44 белка, соответствующие аналитическим фильтрам, которые находились под повышающей или понижающей регуляцией при sPTB в сравнении с полносрочными контролями.
Пример 8. Механистические идеи для сывороточных протеомных биологических маркеров, предсказывающих самопроизвольные преждевременные роды
[00382] Этот пример демонстрирует, что, поскольку экспрессия конкретного белка меняется динамически на всем протяжении беременности, эффективность биологического маркера значительно варьирует на протяжении GA. Дифференциально экспрессированные белки имеют функции в метаболизме стероидов, развитии плаценты, иммунологической толерантности, ангиогенезе и продолжении беременности. Фиг. 55, 57-59. Эти различия в профилях белков, наблюдаемые при sPTB, отражают ослабленные переходы при развитии в фето-плацентарном компартменте во время второго триместра.
[00383] В кратком изложении, цель исследования, описанного в этом примере, состояла в том, чтобы сформировать идею о физиологической основе для связи биологических маркеров с предсказанием самопроизвольных преждевременных родов (sPTB).
[00384] План исследования
[00385] Пути, такие как воспаление, инфекция и кровотечение, вовлечены в этиологию преждевременных родов. Однако мало известно о том, какие белки подвержены измерению в крови и когда при беременности они нарушены. Чтобы ответить на эти вопросы, авторы изобретения создавали LC-MS (MRM) анализ 148 белков из нескольких путей и анализировали образцы сыворотки из 17-25 недель гестационного возраста (GA) от 312 женщин (104 случая sPTB, 208 полносрочных контролей).
[00386] В кратком изложении, образцы сыворотки истощали по белкам с высоким относительным содержанием, расщепляли трипсином и обогащали сильно меченными стабильными изотопами стандартными (SIS) пептидами для почти всех белков. SIS пептиды использовали для нормализации посредством создания коэффициентов ответа, где площадь пика пептидного фрагментарного иона (т. е. перехода), измеренную в сыворотке, делили на таковую для соответствующего SIS перехода. Коэффициенты ответа из данных MRM площади пика анализировали с помощью иерархической кластеризации, критериев Стьюдента и зависимости от GA.
[00387] Как показано на фиг. 53, несколько пептидов хорошо коррелируют с одним и тем же белком. Отдельные ветви (сгруппированные по цвету) соответствуют идентифицируемым функциональным категориям, таким как: белки острой фазы, аполипопротеины и известные специфичные белки беременности. Очевидны белковые комплексы, важные для биологии воспроизводства, такие как: PAPP1:PRG2, INHBE:INHBC и IGF2:IBP3:ALS. Эти качественные оценки и выделенные зависимости валидируют высоко мультиплексный MRM-MS анализ, описанный в этой заявке, для использования в исследовании биологии беременности и исследовании анализируемых веществ, предсказывающих sPTB.
[00388] На фиг. 54 представлены дифференциально экспрессированные белки, которые выполняют функцию во взаимодействиях со внеклеточным матриксом. TENX активирует латентный TGF-b и локализован в строме плода и матери в точках перехода при дифференцировке цитотрофобласта. Alcaraz Alcaraz, L., et al. 2014 J. Cell Biol. 205(3) 409-428; Damsky, C., et al. 1992 J. Clin. Invest. 89(1) 210-222. Сниженные уровни TENX в сыворотке при sPTB указывают на дефекты кровеносных сосудов или сниженную активность TGF-b в плаценте. NCAM1(CD56) высоко экспрессирован на нервных клетках и естественных киллерных клетках. NCAM1 также экспрессируют эндоваскулярные трофобласты, но он снижен или отсутствует в PE плацентах. Red-Horse, K., et al. 2004 J. Clin. Invest. 114:744-754. Инвертированные уровни NCAM1 в сыворотке в случаях sPTB могут отражать слабое ремоделирование спиральной артерии и/или дефектную иммунорегуляцию. CHL1 гомологичен NCAM1 и направляет интегрин-опосредованную клеточную миграцию. BGH3(TGFBI), молекула клеточной адгезии, экспрессирована в эндотелиальных клетках сосудов и ингибирует ангиогенез через специфичные взаимодействия с интегрином αv/β3. Son, H-N., et al. 2013 Biochimica et Biophysica Acta 1833(10) 2378-2388. Повышенный TGFBI в случаях sPTB может указывать на сниженный плацентарный ангиогенез.
[00389] На фиг. 55 представлены кинетические графики дифференциально экспрессированных белков с функциями в пути IGF-2, которые показывают максимальное разделение в 18 недель. IGF2 стимулирует пролиферацию, дифференцировку и эндометриальную инвазию вневорсинчатыми трофобластами при ранней беременности. IBP4 связывает и модулирует биодоступность IGF2 на трансплацентраном барьере. Повышенный IBP4 и сниженный IGF2 во время первого триместра коррелируют с IUGR и SGA, соответственно. Qiu, Q., et al. 2012 J. Clin. Endocrino.l Metab. 97(8):E1429-39; Demetriou, C., et al. 2014 PLOS 9(1): e85454. PAPP1 представляет собой специфичную протеазу плаценты, которая расщепляет IBP4 и высвобождает активный IGF2. Низкие уровни PAPP1 в сыворотке при ранней беременности связаны с IUGR, PE и PTB. Huynh, L., et al. 2014 Canadian Family Physician 60(10) 899-903. PRG2 (proMBP) экспрессирован в плаценте и ковалентно связывает и инактивирует PAPP1. Неактивный комплекс PRG2:PAPP1 циркулирует в материнской сыворотке. Huynh, L., et al. 2014 Canadian Family Physician 60(10) 899-903. Нарушенная регуляция пути находится в соответствии с дефектной активностью IGF2 в случаях sPTB, что может вести к ненормальному формированию плаценты. На фиг. 56A представлена схема динамической регуляции и биодоступности указанных выше белков во время sPTB.
[00390] На фиг. 56B представлена схема внутриклеточных сигналов, предпочтительно активируемых посредством связывания инсулина с IR-B и посредством связывания инсулина и IGF с IR-A или IGF1R. Belfiore and Malaguarnera, Endocrine-Related Cancer (2011) 18 R125-R147. Активация IR-A и IGF1R с помощью инсулина и IGF ведет к преобладанию сигналов роста и пролиферации через фосфорилирование белков IRS1/2 и Shc. Активация Shc ведет к рекрутированию комплекса Grb2/Sos с последующей активацией Ras/Raf/MEK1 и Erk1/2. Эта последняя киназа перемещается в ядро и вызывает транскрипцию нескольких генов, вовлеченных в клеточную пролиферацию и выживаемость. Фосфорилирование IRS1/2 вызывает активацию пути PI3K/PDK1/AKT. Помимо ее роли в метаболических эффектах, AKT ведет к активации эффекторов, вовлеченных в контроль апоптоза и выживаемости (BAD, Mdm2, FKHR, NFkB и JNK) и синтез белка и клеточный рост (mTOR).
[00391] На фиг. 57 представлены кинетические графики дифференциально экспрессированных белков с функциями в равновесии метаболических гормонов. Связывающий половые гормоны глобулин (SHBG), плацентарный белок, повышается во время беременности и определяет биодоступность и метаболизм половых стероидных гормонов. Сниженные уровни SHBG ведут к более высоким уровням свободных андрогенов и эстрогенов. Свободные андрогены могут превращаться в эстрогены через активность плацентарной ароматазы. Прогестерон, противодействующий активности эстрогенов, ускоряет беременность/родовой акт. Эстроген индуцирует связывающий тироксин глобулин (THBG), который возрастает ~2,5-кратно к середние беременности. Повышенные уровни THBG в сыворотке в случаях sPTB могут вести к сниженному свободному тиреоидному гормону. Гипотиреоидизм при беременности связан с повышенным риском выкидыша и преждевременных родов. Stagnaro-Green A. and Pearce E. 2012 Nat. Rev. Endocrinol. 8(11):650-8. Эстроген увеличивает ангиотензиноген ~3-кратно к середине беременности, чтобы стимулировать ~40% увеличение объема плазмы. Повышающая регуляция ANGT может вести к гипертензии беременных, состоянию, связанному с повышенным риском sPTB.
[00392] На фиг. 58 представлены кинетические графики дифференциально экспрессированных белков с функциями в ангиогенезе. TIE1, ингибиторный корецептор рецептора ангиопоэтина TIE2, блокирует способность Ang-2 стимулировать ангиогенез. Seegar, T., et al. 2010 Mol. Cell. 37(5): 643-655. Фактор пигментного эпителия (PEDF), антиангиогенезный фактор, экспрессированный в плаценте, стимулирует расщепление и инактивацию VEGFR-1 γ-секретазой.10 Катепсин D (CATD) расщепляет пролактин для того, чтобы создавать вазоингибины, которые ингибируют ангиогенез. Повышенные CATD и вазоингибины сыворотки связаны с предэклампсией. Nakajima, R., et al. 2015 Hypertension Research 38, 899-901. Богатый лейцином α-2-гликопротеин (LRG1/A2GL) способствует передаче сигнала TGF-β через связывание корецептора, эндоглина. TGF-β активирует митогенез клеток эндотелия и ангиогенез посредством пути передачи сигнала Smad1/5/8. Wang, X., et al. 2013 Nature 499(7458). PSG3 индуцирует противовоспалительные цитокины из моноцитов и макрофагов и стимулирует ангиогенез через связывание TGF-β. Низкие уровни PSG связаны с IUGR. Moore, T., and Dveksler, G. 2014 Int. J. Dev. Biol. 58: 273-280. ENPP2 (аутотаксин), эктофермент с активностью лизофосфолипазы D, образует лизофосфатидную кислоту (LPA). LPA действует на плацентарные рецепторы для того, чтобы стимулировать ангиогенез и хемотаксис естественных киллерных клеток и моноцитов. Уровни аутотаксина снижены в случае PIH и раннего начала PE. Chen, S-U., et al. 2010 Endocrinology 151(1):369-379.
[00393] На фиг. 59 представлены кинетические графики дифференциально экспрессированных белков с функциями во врожденном иммунитете. LBP презентирует бактериальные LPS Toll-подобному рецептору 4 через его корецептор CD14, чтобы индуцировать воспалительную реакцию по пути врожденного иммунитета. Фетуин-A (α-2-HS-гликопротеин) представляет собой белок-переносчик для жирных кислот в крови, и комплекс FetA-FA может связывать и активировать рецептор TLR4. Pal, D., et al. 2012 Nature Med. 18(8):1279-85.
[00394] На фиг. 60 представлены кинетические графики дифференциально экспрессированных белков с функциями в коагуляции.
[00395] На фиг. 61 представлены кинетические графики дифференциально экспрессированных сывороточных/секретируемых белков.
[00396] На фиг. 62 представлены кинетические графики дифференциально экспрессированных PSG/IBP.
[00397] На фиг. 63 представлены кинетические графики дифференциально экспрессированных ECM/белков клеточной поверхности.
[00398] На фиг. 64 представлены кинетические графики дифференциально экспрессированных белков комплемента/острой фазы-1.
[00399] На фиг. 65 представлены кинетические графики дифференциально экспрессированных белков комплемента/острой фазы-2.
[00400] На фиг. 66 представлены кинетические графики дифференциально экспрессированных белков комплемента/острой фазы-3.
[00401] Фиг. 67 представлены кинетические графики дифференциально экспрессированных белков комплемента/острой фазы-4.
Пример 9. Кинетический анализ SDT4/SV4
[00402] в этом примере предоставлен кинетический анализ для всех анализируемых веществ, изначально приведенных в примере 1, выше, с использованием данных от 17 недель 0 суток до 28 недель 6 суток.
[00403] Для фиг. 68-85 усредненные удельные соотношения для каждого перехода пептида нанесены на график с использованием пакета R ggplot2 в зависимости от GABD, используя функцию сглаживания средней (интервал=+/- 10 суток). Графики отличаются отдельными построениями для случая и контроля с использованием двух различных порогов гестационного возраста при рождении (<37 0/7 в сравнении с≥37 0/7 неделями и <35 0/7 в сравнении с≥35 0/7 неделями). В названии графика отображено короткое название белка, подчеркивание и пептидная последовательность. Для названий последовательности анализируемых веществ могли быть усечены, чтобы помещаться на графике.
[00404] Кинетические анализы, приведенные в качестве примера в настоящем описании, служат нескольким целям. Эти анализы демонстрируют, меняются ли уровни анализируемых веществ во время беременности и в каком направлении, меняются ли они различно для случаев и контролей и иллюстрируют диагностические различия в качестве функции гестационного возраста. В некоторых случаях, диагностический сигнал находится в узком диапазоне гестационного возраста и возрастает или снижается с течением времени. Геометрическая форма кинетических графиков также предоставляет визуальное руководство для выбора белков, которые хорошо образуют пары в обращениях.
[00405] Анализируемые вещества, для которых обнаружено, что они демонстрируют значительное разделение случаев и контролей в раннем интервале, например, совокупность образцов между 18 и 20 неделями гестационного возраста, включают, например, AFAM, B2M, CATD, CAH1, C1QB, C1S, F13A, GELS, FETUA, HEMO, LBP, PEDF, PEPD, PLMN, PRG2, SHBG, TENX, THRB и VCAM1. Анализируемые вещества, для которых обнаружено, что они демонстрируют значительное разделение случаев и контролей в позднем интервале, например, совокупность образцов между 26 и 28 неделями гестационного возраста, включают, например, ITIH4, HEP2, IBP3, IGF2, KNG1, PSG11, PZP, VASN и VTDB. Разделение случаев и контролей улучшали с использованием порога > 35 0/7 в сравнении с≤35 0/7 недель в сравнении с > 37 0/7 в сравнении с≤37 0/7 недель, как видно для анализируемых веществ, включая, например, AFAM, APOH, CAH1, CATD, CD14, CLUS, CRIS3, F13B, IBP6, ITIH4, LYAM1, PGRP2, PRDX, PSG2, PTGDS, SHBG и SPRL1. Обнаружено, что многие воспалительные и иммуномодуляторные молекулы демонстрируют усовершенствованное разделение при использовании более раннего порога гестационного возраста при рождении. Специалист в данной области примет во внимание, что какие-либо из анализируемых веществ, демонстрирующих значительное разделение между случаями и контролями, которые приведены на сопроводительных фиг. для заданного временного интервала, являются кандидатами для использования в парах обращения по настоящему изобретению, в качестве отдельного биологического маркера или в качестве части панели биологических маркеров анализируемых веществ.
[00406] Наконец, кинетические графики для анализируемых веществ, на которых отсутствует разница между случаями и контролями, но которые демонстрируют изменения интенсивности анализируемых веществ на протяжении беременности, можно использовать для хронологии беременности в соответствии со способами по изобретению. Эти анализируемые вещества, также упоминаемые в настоящем описании как «хронологические белки», можно использовать для того, чтобы датировать беременность в отсутствие других способов датирования или в сочетании с ними (например, дата последнего менструального цикла, ультразвуковое датирование). В таблице 60 предоставлен список хронологических белков, которые можно использовать для хронологии беременности по изобретению.
Пример 10. Исследование 2 Анализ случаев sPTB
[00407] В этом примере описан анализ всех предварительно анализированных случаев sPTB, как описано в предшествующих примерах, соответствующие им контроли (2 на каждый случай) и 2 новых контроля. Этот анализ, описанный в этом примере, расширял коммерческий интервал взятия крови за пределы недель 19 и 20, создавал дополнительные данные в отношении предсказания sPTB <35 недель на основании большего числа образцов из всех предыдущих примеров, вел к исследованию новых анализируемых веществ и обращений, определял белки молекулярных часов, прояснял пороги риска и формировал точные валидационные требования для будущих клинических исследований.
[00408] Способы обработки образцов
[00409] Разрабатывали стандартный протокол, который определяет проведение клинического исследования протеомной оценки преждевременного риска (PAPR). Этот протокол также определяет то, что образцы и клиническую информацию можно использовать для исследования других осложнений беременности. Образцы получали от женщин в 11 местах, одобренных Internal Review Board (IRB), по всем Соединенным Штатам. После предоставления информированного согласия, получали образцы сыворотки и плазмы, а также уместную информацию, касающуюся демографических характеристик пациентов, прошлого медицинского анамнеза и анамнеза беременности, анаменеза текущей беременности и сопутствующих медикаментов. После родоразрешения, собирали данные, касающиеся состояний и осложнений у матерей и младенцев. Образцы сыворотки и плазмы обрабатывали в соответствии с протоколом, который требует стандартизованного охлажденного центрифугирования, разделения образцов на аликвоты в 0,5 мл криофлаконах с двухмерным штриховым кодом и последующей заморозки при -80°C.
[00410] После родоразрешения, случаи преждевременных родов индивидуально просматривали для того, чтобы определять их статус в качестве самопроизвольных преждевременных родов или медицински показанных преждевременных родов. Для этого анализа использовали только самопроизвольные случаи преждевременных родов. Для исследования биологических маркеров преждевременных родов генерировали LC-MS данные для 413 образцов (82 случая sPTB, 331 полносрочный контроль), покрывающих гестационные возрасты от 17 0/7 до 21 6/7 недель, причем каждому преждевременному образцу соответствовало 4 полносрочных контроля по гестационному возрасту при взятии крови. Каждые сутки гестационного возраста в пределах от 17 0/7 до 21 6/7 недель включали по меньшей мере один случай sPTB (и соответствующие полносрочные контроли), за исключением одних суток. Выбирали 4 полносрочных контроля со взятиями крови в эти сутки. Один полносрочный контроль в исследовании, для которого не удалось провести лабораторный анализ, не анализировали повторно.
[00411] Образцы сыворотки впоследствии истощали по белкам с высоким относительным содержанием, используя Human 14 Multiple Affinity Removal System (MARS-14), которая удаляет 14 наиболее обильных белков. Равные объемы каждого клинического образца или повторений двух объединенных сывороток для контроля качества разводили буфером для колонки и фильтровали для того, чтобы удалять преципитаты. Фильтрованные образцы истощали с использованием колонки MARS-14 (4,6×100 мм, Agilent Technologies). Образцы охлаждали до 4°C в автоматическом пробоотборнике, колонку для истощения прогоняли при комнатной температуре и собранные фракции хранили при 4°C до последующего анализа. Несвязанные фракции собирали для последующего анализа.
[00412] Истощенные образцы сыворотки восстанавливали дитиотреитолом, алкилировали с использованием йодацетамида и затем расщепляли трипсином. После расщепления трипсином образцы обогащали объединенными стабильными изотопными стандартами в концентрациях, которые приближались к концентрации анализируемого вещества суррогатного пептида. SIS обогащенные образцы смешивали и делили на два равных объема. Каждую часть помещали на хранение при -80°C до тех пор, пока не были готовы продолжать рабочий процесс. Одну замороженную часть из каждого образца извлекали из хранения при -80°C, оставляли оттаивать и затем обессоливали на планшете C18 для твердофазного экстрагирования (Empore, 3M). Элюированные пептиды лиофилизировали досуха. Лиофилизированные образцы ресолюбилизировали в растворе для восстановления, содержащем внутренние стандарты только для осуществления мониторинга качества стадии LC-MS (IS Recon).
[00413] Полностью обработанные образцы анализировали с использованием способа динамического мониторинга нескольких реакций (dMRM). Пептиды разделяли на колонке Poroshell EC-C18 2,1×100 мм с размером частицы 2,7 μ при скорости потока 0,4 мл/мин, используя Agilent 1290 UPLC, и элюировали с использованием градиента ацетонитрила в тройном квадрупольном масс-спектрометре Agilent 6490 с электрораспылительным источником, работающим в режиме положительных ионов. В dMRM анализе измеряли 442 перехода, которые соответствуют 119 пептидам и 77 белкам, играющим как диагностические, так и качественные роли. Хроматографические пики интегрировали с использованием программного обеспечения MassHunter Quantitative Analysis (Agilent Technologies). Приведены соотношения площади хроматографического пика анализируемого вещества суррогатного пептида и соответствующей площади SIS хроматографического пика.
[00414] Сводка по белкам, пептидам и переходам для сывороточных анализируемых веществ, SIS переходам и стандартам IS Recon, которые измеряли в способе dMRM, представлена в таблице 21. Истощенные MARS-14 белки обозначают анализируемые вещества, на которые направлена иммунная колонка для истощения MARS-14 и которые измеряют для целей контроля качества. Количественные переходы используют для относительных коэффициентов ответа и качественные переходы служат целям контроля качества. Знак сноски (*) обозначает изменения названий. CSH обозначает, что пептид соответствует как CSH1, так и CSH2. HLAG здесь обозначают как HLACI, поскольку пептид консервативен в нескольких изотипах класса I HLA. LYAM3 здесь обозначают как LYAM1, поскольку, хотя пептидная последовательность присутствует в каждой, ее получают только посредством расщепления трипсином из LYAM1. SOM2 здесь обозначают как SOM2.CSH, поскольку пептиды специфичны как для SOM2, так и для CSH.
[00415] Выбор значимых белков и обращений
[00416] Для каждого анализируемого вещества в каждом из двухнедельного и трехнедельного перекрывающегося интервала, с ограничением BMI и без него и с двумя определениями SPTB (37/37 и 35/35), вычисляли значение кратности изменения, которое обозначает, что среднее образцов для случаев sPTB было выше или ниже среднего полносрочных контрольных образцов. В таблицах 22 и 23 представлена AUROC белков/переходов для двухнедельных интервалов гестационного возраст с перекрытием в одну неделю (например, 119-132 относится к суткам 119-132 беременности, которые равны неделям беременности 17 и 18). Эффективность в каждом двухнедельном интервале приведена для двух различных порогов случай-контроль (<37 0/7 в сравнении с≥37 0/7, <35 0/7 в сравнении с≥35 0/7) и с (rBMI) и без (aBMI) BMI-стратификации. В таблицах 24 и 25 представлена AUROC белков/переходов для трехнедельных интервалов беременности с перекрытием в две недели (показаны в сутках, например, «119-139» относится к суткам 119-139 беременности, которые соответствуют неделям беременности 17, 18 и 19). Эффективность в каждом трехнедельном интервале приведена для двух различных порогов случай-контроль (<37 0/7 в сравнении с≥37 0/7, <35 0/7 в сравнении с≥35 0/7) и с (rBMI) и без (aBMI) BMI-стратификации.
[00417] На фиг. с 86 до 95 представлены кинетические графики различных переходов пептидов для случая в сравнении с контролем с использованием порога гестационного возраста при рождении <37 0/7 в сравнении с≥37 0/7 недель. На фиг. с 96 до 105 представлены кинетические графики различных переходов пептидов для случая в сравнении с контролем с использованием порога гестационного возраста при рождении <35 0/7 в сравнении с≥35 0/7 недель. В кратком изложении, усредненные удельные соотношения для каждого перехода пептида нанесены на график с использованием пакета R ggplot2 в зависимости от GABD, используя функцию сглаживания средней (интервал=+/- 10 суток). Графики отличаются отдельными построениями для случая в сравнении с контролем с использованием двух различных порогов гестационного возраста при рождении (<37 0/7 в сравнении с≥37 0/7 недель и <35 0/7 в сравнении с≥35 0/7 недель). В названии графика отображено короткое название белка, подчеркивание и пептидная последовательность. Последовательности анализируемых веществ могли быть усечены для названий, чтобы помещаться на графике.
[00418] На основании значения кратности изменения, которое обозначает, что среднее образцов для случаев sPTB выше или ниже среднего полносрочных контрольных образцов, каждое анализируемое вещество помечали как находящееся под повышающей или понижающей регуляцией для каждой из комбинаций (т. е. перекрывающийся 2- или 3-недельный интервал, ограничение BMI и определение SPTB), и если анализируемое вещество имело большинство комбинаций, помеченных как находящиеся под повышающей регуляцией, его называли анализируемым веществом в целом под повышающей регуляцией, и наоборот. Это показано в таблице 26.
[00419] На основании этих присваиваний повышающей и понижающей регуляции (55 под повышающей регуляцией и 30 под понижающей регуляцией), обращения создавали посредством деления каждого из значений удельных соотношений анализируемых веществ под повышающей регуляцией на таковые для анализируемых веществ под понижающей регуляцией и получения натурального логарифм результата. Это давало 1650 обращений (55×30=1650). Для каждого обращения вычисляли площадь под ROC-кривой (AUCROC), обозначающую разделение SPTB и TERM0, и p-значение, показывающее, если значение AUCROC значительно отличается от AUCROC=0,5 (т. е. нет значимого разделения SPTB и TERM). Эффективность каждого обращения сводили в таблицу для различных условий (например, интервалы беременности, с или без ограничения BMI и два порога sPTB), для обращений с AUCROC > 0,6 и p-значением < 0,05. В таблицах с 27 до 42 представлена эффективность классификации по обращениям для недель беременности 17 и 18. В таблице с 47 до 58 представлена эффективность классификации по обращениям для недель беременности 17, 18 и 19. В таблицах с 43 до 46 представлена эффективность классификации по обращениям для недель беременности с 17 до 21. Дополнительные обращения с потенциальной значимостью представлены в таблице 59.
[00420] Также продемонстрирована усовершенствованная эффективность предикторов, сформированных из больше чем одного обращения (недели 17-21). В кратком изложении, комбинировали обращения, которые давали высокую предсказательную эффективность, или раньше (например, недели 17-19) или позже (например, недели 19-21) в этом диапазоне гестационного возраста, и оценивали эффективность предикторов, сформированных из комбинаций (SumLog) нескольких обращений для всего диапазона взятия крови. Это показано в таблице 61. Оценку предиктора получали суммированием логарифмических значений индивидуального обращения (SumLog), но специалист в данной области может выбрать другие модели (например, логистическую регрессию). Также предусмотрено применение этого подхода с несколькими обращениями к комбинациям обращений, специфичных для преждевременного разрыва околоплодных оболочек (PPROM), в сравнении с преждевременными сокращениями матки в отсутствие PPROM (PTL), полом плода и числа беременностей. Кроме того, предусмотрено, что предиктор может содержать индикаторную переменную, которая выбирает поднабор обращений, подлежащих использованию, принимая во внимание сведения о периоде взятия крови, поле плода или числе беременностей.
[00421] На фиг. 110 представлены зависимости между оценкой предиктора (ln IBP4/SHBG) и скорректированным по болезненности относительным риском sPTB (положительное прогностическое значение), используя порог <37 0/7 недель в сравнении с≥37 0/7 недель беременности. Образцы брали между 19 1/7 неделями и 20 6/7 неделями с BMI >22≤37. Относительный риск возрастает с увеличением оценки предиктора от фоновой величины 7,3% (риск sPTB в усредненной популяции при одноплодных беременностях) приблизительно до 50%. Наложена скрининговая кривая положительной величины для всех оценчных порогов. Доверительные области (серые) вычисляли, исходя из биномиально распределенных наблюдений и аппроксимации распределения ошибки к нормальному распределению. Распределение образцов по оценке классификатора представлено с помощью линейчатой диаграммы в соответствии с цветовой схемой в легенде на фиг.
[00422] На фиг. 111 представлена зависимость между оценкой предиктора (ln IBP4/SHBG) и скорректированным по болезненности относительным риском sPTB (положительное прогностическое значение), используя порог <35 0/7 недель в сравнении с≥35 0/7 недель беременности. Образцы брали между 19 1/7 неделями и 20 6/7 неделями. Относительный риск возрастает с увеличыением оценки предиктора от фоновой величины 4,4% (риск sPTB в усредненной популяции (<35) при одноплодных беременностях) приблизительно до 50%. Наложена скрининговая кривая положительной величины для всех оценчных порогов. Доверительные области (серые) вычисляли, исходя из биномиально распределенных наблюдений и аппроксимации распределения ошибки к нормальному распределению. Распределение образцов по оценке классификатора представлено с помощью линейчатой диаграммы в соответствии с цветовой схемой в легенде на фиг.
[00423] Клинические наблюдения: sPTB, PPROM и PTL
[00424] Эффективность обращения (недели GABD 17-21) оценивали независимо для двух различных фенотипов sPTB, PPROM и PTL. PPROM более часто встречается рано и связан с инфекцией или воспалением. PTL может возникать позже, и в целом его считают менее тяжелым фенотипом. Имели место более значимые обращения и с более высокой эффективностью для PPROM и эти обращения заполнены белками, о которых известно, что они вовлечены в воспаление и инфекцию. Предусмотрен выбор обращений для создания способов независимого тестирования PPROM и PTL или для максимизации общей эффективности с использованием комбинации из больше чем одного обращения в одном предикторе. В анализе, представленном в таблицах с 61 до 64, необходимо AUC > 0,65 и p <0,05 для PPROM или PTL.
[00425] В таблице 61 представлена AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, без BMI-стратификации, отдельно для PPROM и PTL. В таблице 62 представлена AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, с BMI-стратификацией (>22≤37), отдельно для PPROM и PTL. В таблице 63 представлена AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, без BMI-стратификации, отдельно для PPROM и PTL. В таблице 64 представлена AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, с BMI-стратификацией (>22≤37), отдельно для PPROM и PTL.
[00426] Также определяли наиболее эффективные анализируемые вещества для PTL и наиболее эффективные анализируемые вещества для PPROM для недель GABD 19-20, и из наиболее эффективных конструировали несколько обращений. IBP4 присутствует в качестве высоко эффективного как в PTL, так и в PPROM, что допускает его полезность в целом для sPTB. В таблице 76 перечислены AUROC переходов для недель беременности от 19 1/7 до 20 6/7 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, без BMI-стратификации, для PTL. В таблице 77 перечислены AUROC переходов для недель беременности от 19 1/7 до 20 6/7 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, без BMI-стратификации, для PPROM. На фиг. 108 в качестве примера приведены обращения с хорошей эффективностью в недели 19-20 при PTL. На фиг. 109 приведены в качестве примера обращения с хорошой эффективностью в недели 19-20 при PPROM.
[00427] Клинические наблюдения: первая беременность и повторная беременность
[00428] Эффективность обращения (недели 17-21) дополнительно оценивали независимо для двух различных фенотипов sPTB, первой беременности и повторной беременности. В таблицах 65-68 наиболее эффективные обращения (недели 17-21) представлены отдельно для субъектов с первой беременностью («матери в первый раз») и повторной беременностью. «Матери в первый раз» больше всего нуждаются в тесте для предсказания вероятности PTB, поскольку они не имеют анамнеза беременности для врачей, чтобы определять/оценивать риск. Эти результаты делают тест независимым для этих двух групп, или позволяют комбинировать высоко эффективные обращения в одном классификаторе для того, чтобы предсказать риск для тех и других. В анализе, представленном в таблицах 65-68, необходимо AUC > 0,65 и p <0,05 для первой беременности или повторной беременности.
[00429] В таблице 65 представлена AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, без BMI-стратификации, отдельно для первой беременности и повторной беременности. В таблице 66 представлена AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, с BMI-стратификацией (>22≤37), отдельно для первой беременности и повторной беременности. В таблице 67 представлена AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, без BMI-стратификации, отдельно для первой беременности и повторной беременности. В таблице 68 представлена AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, с BMI-стратификацией (>22≤37), отдельно для первой беременности и повторной беременности.
[00430] Клинические наблюдения: пол плода
[00431] Эффективность обращения (недели 17-21) дополнительно оценивали независимо для субъектов, беременных плодом мужского пола в сравнении с плодом женского пола. Обнаружено, что некоторые обращения обладают предсказательной эффективностью, специфичной для пола плода. На фиг. 106 показаны различия анализируемых веществ, специфичных к полу плода, IBP4 и SHBG и оценок (IBP4/SHBG). IBP4 значимо выше у субъектов с плодами мужского пола. Эффективность обращения остается сравнимой для недель гестационного возраста 19-21 без BMI-стратификации (фиг. 106). Дополнительно, в PAPR клиническом исследовании обнаружено, что плоды мужского пола имеют повышенный риск sPTB с p-значением 0,0002 и отношением шансов 1,6. Наконец, пол плода можно включать в предиктор (например, значение обращения плюс пол плода). В анализе, представленном в таблицах 69-72, необходимо AUC > 0,65 и p <0,05 для плодов мужского или женского пола.
[00432] В таблице 69 представлена AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, без BMI-стратификации, отдельно по полу плода. В таблице 70 представлена AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, с BMI-стратификацией (>22≤37), отдельно по полу плода. В таблице 71 представлена AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, без BMI-стратификации, отдельно по полу плода. В таблице 72 представлена AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, с BMI-стратификацией (>22≤37), отдельно по полу плода.
Пример 11. Корреляция данных масс-спектрометрии и иммунологического анализа
[00433] Этот пример демонстрирует реализацию иммунологических анализов с использованием платформы MSD (например, MSD данных, коррелирующих с коммерческими данными ELISA и данными MS для IBP4 и SHBG).
[00434] Материалы
[00435] Использовали следующие антитела: связывающий половые гормоны глобулин (Biospacific №№ по каталогу 6002-100051 и 6001-100050; R&D Systems №№ по каталогу MAB2656 и AF2656), IGFBP-4 (Ansh №№ по каталогу AB-308-AI039 и AB-308-AI042). Белки SHBG из Origene (№ по каталогу TP328307), Biospacific (№ по каталогу J65200), NIBSC (код 95/560) и R&D Systems (доступно только в качестве части комплекта ELISA SHBG) тестировали в качестве калибратора. Рекомбинантный IGFBP-4 человека (Ansh, № по каталогу AG-308-AI050) использовали в качестве калибратора.
[00436] Создание растворов индивидуальных антител, сопряженных с U-PLEX
[00437] Каждое биотинилированное антитело разводили до 10 мкг/мл в Diluent 100 до конечного объема≥200 мкл. Затем биотинилированное антитело добавляли в 300 мкл прилагаемого U-PLEX Linker. (Для каждого биотинилированного антитела использовали отличающийся линкер). Образцы энергично перемешивали и инкубировали при комнатной температуре в течение 30 минут. Stop Solution (200 мкл) добавляли в каждую пробирку. Пробирки энергично перемешивали и инкубировали при комнатной температуре в течение 30 минут.
[00438] Получение Multiplex Coating Solution
[00439] Раствор каждого U-PLEX-сопряженного антителоа (600 мкл) объединяли в одной пробирке и перемешивали на вортексе. При объединении меньше чем 10 антител, объем раствора доводили вплоть до 6 мл с использованием Stop Solution, чтобы получать конечную концентрацию 1×. Следует отметить, что в этих экспериментах имело место только одно антитело на лунку.
[00440] Покрывание планшета U-PLEX.
[00441] В каждую лунку добавляли Multiplex Coating Solution (50 мкл). Планшеты запечатывали адгезивным запечатывающим средством для планшетов и инкубировали при комнатной температуре в течение 1 часа или при 2-8˚C в течение ночи, при встряхивании приблизительно на 700 об./мин. После промывания 3 раза с использованием по меньшей мере 150 мкл 1× MSD буфера для промывания, планшеты были готовы для использования.
[00442] Анализ образцов
[00443] Аликвоты, 50 мкл, образца или калибратор добавляли в каждую лунку. Планшет запечатывали и инкубировали при комнатной температуре в течение 1 часа при встряхивании приблизительно на 700 об./мин. Затем планшет промывали 3 раза с использованием по меньшей мере 150 мкл 1× MSD буфера для промывания. В каждую лунку добавляли раствор идентифицирующего антитела, 50 мкл. После запечатывания, планшет инкубировали при комнатной температуре в течение 1 часа при встряхивании приблизительно на 700 об./мин. Планшет промывали 3 раза с использованием по меньшей мере 150 мкл 1× MSD буфера для промывания. После добавления 150 мкл 2× Read Buffer в каждую лунку, планшет незамедлительно считывали на приборе MSD.
[00444] Скрининг SHBG антител и калибратора
[00445] Все антитела тестировали в обеих ориентациях иммобилизованных и идентифицирующих антител, все попарные комбинации. Получали иммобилизованные антитела 10 мкг/мл, сопряженные с U-PLEX Linker, и покрывали ими планшет U-PLEX. Калибратор SHBG R&D Systems разводили в Diluent 43 для того, чтобы создавать 7-точечную калибровочную кривую с пустой пробой из разбавителя для анализа. Образцы разводили следующим образом в Diluent 43 и тестировали в анализах: образцы Sera с «высоким» и «низким» SHBG: 100- и 500-кратные разведения и Sera Pregnant Pool: 100-, 200-, 400-, 800-кратные разведения. Идентифицирующие антитела тестировали в концентрации 1 мкг/мл в Diluent 3. Оценивали калибровочные кривые и связывание с нативным анализируемым веществом в сыворотке. Затем наилучшие пары анализируемых веществ тестировали с использованием калибраторов NIBSC и Biospacific, в разведениях, полученных, как указано выше.
[00446] Скрининг антител к IGFBP-4 и калибратора.
[00447] Два антитела тестировали в обеих ориентациях иммобилизованных и идентифицирующих антител. Получали иммобилизованные антитела 10 мкг/мл, сопряженные с U-PLEX Linker, и покрывали ими планшет U-PLEX. Калибратор IGFBP-4 разводили в Diluent 12 и для того, чтобы создавать 7-точной калибровочной кривой с пустой пробой из разбавителя для анализа. Образцы разводили следующим образом в Diluent 12 и тестировали в анализах: образцы Sera с «высоким» и «низким» IGFBP-4: 5-кратно, Sera Pregnant Pool: 2-кратные разведения от 2 до 64 крат, и 2 индивидуальных образца сывороток человека (MSD образцы): 2-, 4-, 8- и 16-кратно. Тестировали идентифицирующие антитела 1 мкг/мл в Diluent 12. Оценивали калибровочные кривые и связывание с нативным анализируемым веществом в сыворотке.
[00448] Тестирование SHBG и IGFBP-4 с использованием 60 образцов Sera.
[00449] Пару антител 12 выбирали для того, чтобы в двух повторениях измерять SHBG в 60 образцах плазмы из Sera. Для IGFBP-4 выбирали пару 2. Образцы плазмы разводили 1:1000 и 1:20 для SHBG и IGFBP-4, соответственно. Результаты MSD ELISA сравнивали с коммерческими наборами для ELISA и с данными MS-MRM.
[00450] Результаты
[00451] Скрининг антител к SHBG
[00452] Только пара антител 1 (R&D моно, иммобилизованное, поли, обнаружение) давала сильнй сигнал с использованием калибратора Origene, указывая на то, что это калибратор может представлять субпопуляцию эндогенного анализируемого вещества SHBG. Таким образом, дополнительные калибраторы тестировали в последующих исследованиях для того, чтобы идентифицировать калибратор, который работает со всеми парами. Тем не менее, все пары антител распознавали нативное анализируемое вещество в образцах Sera «высокий», «низкий» и объединенных Pregnant. R&D поли AF2656 и Biospacific моно 6001-100050 давали схожую эффективность. Пары 2, 3 и 12 демонстрировали приблизительно линейное титрование при разведении образца (таблица 73). Затем четыре наилучших пары антител тестировали на эффективность с использованием трех дополнительных калибраторов. Хорошие кривые калибраторов получали для 4 наилучших пар для 3 калибраторов (таблица 74). Различия в сигнале могу тбыть, отчасти, обусловлены различиями в присвоенных концентрациях.
[00453] В нижней части показано, что сигналы NIBSC или Biospacific в отношении калибратора R&D варьировали в зависимости от пары антител. Пары 3 и 10 (те же антитела с перевернутой ориентацией иммобилизованного и идентифицирующего антител) имели схожий профиль. Пара 2 давала более слабые сигналы для NIBSC и Biospacific (в сравнении с парой 3, то же иммобилизованное антитело). Пара 12 давала более сильные сигналы для Biospacific и больше чем в 3 раза более сильные сигналы для стандарта NIBSC.
[00454] Скрининг антител к IGFBP-4
[00455] Калибровочная крива пары антител 2 давала в 4-6 раз более специфичные сигналы калибратора и фон по сравнению с парой 1 (таблица 75). Сигналы образцов сыворотки попадали в линейный диапазон для большинства тестированных разведений; объединенные образцы беременных достигали фона при 32- и 64-кратном разведениях. Пара 2 давала в ~12 раз более сильные сигналы для образцов, что вело к различиям в количественном определении в 2-4 раза. CV сигналов в целом составляли <5% для обеих пар.
[00456] Измерение SHBG и IGFBP-4 в 60 образцах сыворотки
[00457] Для SHBG, при 1000-кратном разведении, образцы попадали между стандартами калибраторов 1-3. Медианная измеренная концентрация составляла 58,4 мкг/мл. CV измерений в двух повторениях были небольшими при медианном CV 2,4%. Медианная измеренная концентрация для IGFBP-4 составляла 234 нг/мл и медианный CV между образцами в двух повторениях составлял 2,2%. Как показано на фиг. 107, хорошую корреляцию наблюдали для обоих белков в MSD анализе по сравнению с коммерческими наборами для ELISA и MS-MRM анализом.
[00458] Из приведенного описания очевидно, что вариации и модификации можно выполнять в изобретении, описанном в настоящем описании, чтобы адаптировать его к различным использованиям и условиям. Такие варианты осуществления также входят в объем следующей формулы изобретения.
[00459] Изложение списка элементов в любом определении переменной в настоящем описании включает определения этой переменной в качестве любого одного элемента или комбинации (или подкомбинации) перечисленных элементов. Изложение варианта осуществления в настоящем описании включает этот вариант осуществления в виде любого одного варианта осуществления или в комбинации с какими-либо другими вариантами осуществления или их частями.
[00460] Все патенты и публикации, упомянутые в этом описании, включены в настоящее описание посредством ссылки в той же степени, как если бы каждый независимый патент и публикацию конкретно и индивидуально указывали в качестве включенных по ссылке.
Таблица 1: материнские характеристики и исходы беременности, стратифицированные по времени родоразрешения (sPTB и полносрочные роды)
PAPR
Валидация
PAPR и валидация
Случаи
N (%)
(N=217)
Контроли
N (%)
(N=4,292)
p-значение
Случай
N (%)
(N=81)
Контроль
N (%)
(N=162)
p-значение
p-значение
(случаи)
p-значение
(контроли)
Материнские характеристики
Возраст матери при включении в исследование, годы
0,245
0,239
0,741
0,226
18-22 лет
58 (26,7)
990 (23,1)
22 (27,2)
47 (29,0)
23-27 лет
56 (25,8)
1,222 (28,5)
17 (21,0)
41 (25,3)
28-32 лет
54 (24,9)
1,154 (26,9)
25 (30,9)
34 (21,0)
33-37 лет
31 (14,3)
692 (16,1)
9 (11,1)
30 (18,5)
38 или больше
18 (8,3)
234 (5,5)
8 (9,9)
10 (6,2)
Среднее
28
28
28
28
Медиана
27
27
28
27
Интерквартильный размах
22-32
23-32
21-32
22-32
Индекс массы тела, кг/м2
0,380
0,802
0,630
0,191
Меньше 18,5
10 (4,7)
129 (3,1)
1 (1,3)
2 (1,3)
18,5-24,9
78 (36,8)
1,789 (42,3)
25 (31,3)
55 (34,6)
25,0-29,9
54 (25,5)
1,091 (25,8)
26 (32,5)
46 (28,9)
30,0-34,9
39 (18,4)
617 (15,6)
17 (21,3)
25 (15,7)
35,0-39,9
17 (8,0)
320 (7,6)
6 (7,5)
17 (10,7)
Больше 40,0
14 (6,6)
286 (6,7)
5 (6,3)
14 (8,8)
Среднее
27,8
27,5
28,4
29,1
Медиана
26,5
25,7
27,4
27,8
Интерквартильный размах
22,7-31,8
22,3-31,1
23,6-32,0
23,4-32,4
Уровень образования
<0,0002
0,201
0,711
0,094
Ученая степень
13 (6,0)
461 (10,9)
6 (7,7)
14 (8,7)
Диплом университета
34 (15,8)
701 (16,6)
10 (12,6)
22 (13,8)
Любой колледж
51 (23,7)
936 (22,2)
19 (24,0)
23 (14,4)
Аттестат зрелости/эквивалент
46 (21,4)
1,032 (24,5)
16 (20,2)
50 (31,3)
Любая средняя школа
53 (24,6)
774 (18,4)
25 (31,6)
36 (22,5)
9 классов или меньше
12 (5,8)
292 (6,9)
3 (3,8)
14 (8,7)
Другое
6 (2,8)
23 (0,6)
0
1 (0,6)
Этническая принадлежность
0,157
0,035
0,226
0,003
Испанцы или латино
89 (41,0)
1,557 (36,3)
27 (33,3)
77 (47,5)
Не испанцы или латино
128 (59,0)
2,735 (63,7)
54 (66,7)
85 (52,5)
Раса
0,887
0,811
0,953
0,071
Американские индейцы/коренные жители Аляски
1 (0,5)
29 (0,7)
0
2 (1,2)
Азиатская
4 (1,8)
131 (3,1)
1 (1,2)
1 (0,6)
Черная или афро-американцы
45 (20,7)
838 (19,5)
19 (23,5)
37 (22,8)
Коренные гавайцы или другие обитатели тихоокеанских островов
0
12 (0,30)
0
2 (1,2)
Белая
156 (71,9)
3,101 (72,3)
58 (71,6)
114 (70,4)
Другое
11 (5,1)
193 (4,5)
3 (3,7)
6 (3,7)
Акушерские характеристики
Первая беременность
64 (29,5)
1,212 (28,2)
0,689
27 (33,3)
39 (24,1)
0,126
0,522
0,247
Повторная беременность
153 (70,5)
3,080 (71,8)
54 (66,7)
123 (75,9)
Число предшествующих полносрочных родоразрешений
0,007
0,326
0,816
0,881
1 или больше
113 (73,8)
2,538 (82,4)
40 (74,5)
102 (82,9)
Нет
40 (26,2)
542 (17,6)
13 (24,5)
21 (17,1)
Число предшествующих самопроизвольных PTB
<0,0001
0,221
0,337
0,472
1 или больше
35 (22,9)
339 (11,0)
9 (16,7)
11 (8,9)
Нет
118 (77,1)
2,741 (89,0)
45 (83,3)
112 (91,1)
Характеристики образа жизни
Курение
0,412
0,719
0,555
0,283
Да
34 (15,7)
588 (13,7)
15 (18,5)
27 (16,7)
Нет
183 (84,3)
3,704 (86,3)
66 (81,5)
135 (83,3)
Запрещенные средства
0,283
0,628
0,992
0,052
Да
16 (7,4)
242 (5,6)
6 (7,4)
15 (9,3)
Нет
201 (92,6)
4,050 (94,4)
75 (92,6)
147 (90,7)
Алкоголь
0,096
0,628
0,622
0,141
Да
20 (9,2)
273 (6,4)
6 (7,4)
15 (9,3)
Нет
197 (90,8)
4,018 (93,6)
75 (92,6)
147 (90,7)
Употребление алкоголя
0,108
0,592
0,880
0,317
Да (количество не известно)
3 (1,4)
39 (0,9)
0
2 (1,2)
Социально (нерегулярное)
16 (7,4)
230 (5,4)
6 (7,4)
13 (8,0)
Тяжелое (ежедневное)
1 (0,5)
4 (0,09)
0
0
Нет
197 (90,8)
4,018 (93,6)
75 (92,6)
147 (90,7)
Медицинские характеристики
Кровотечение во время беременности после 12 нед.
0,006
0,360
0,785
0,892
Да
21 (9,7)
228 (5,3)
7 (8,6)
9 (5,6)
Нет
196 (90,3)
4,064 (94,7)
74 (91,4)
153 (94,4)
sPTB, самопроизвольные преждевременные роды; PTB, преждевременные роды; N, число субъектов.
Сравнения клинических данных между случаями и контролями осуществляли с использованием критерия хи-квадрат или точного критерия Фишера или критерия Манна-Уитни, в зависимости от ситуации (система SAS 9.4) и R (3.1.0).
Отсутствующие значения исключены из частотных таблиц.
Таблица 2
Граница GA
AUC
Чувствительность
Специфичность
OR (95% CI)
<37 в сравнении с ≥ 37
0,75 (p=0,016)
0,75
0,74
5,04 (1,4-18)
<36 в сравнении с ≥ 36
0,79 (p=0,027)
0,83
0,83
17,33 (2,2-138)
<35 в сравнении с ≥ 35
0,93 (p=0,001)
1,00
0,83
34,47 (1,7-699)
Таблица 3: материнские характеристики и исходы беременности, стратифицированные по времени родоразрешения (sPTD и полносрочные роды)
PAPR исследование
Полная валидационная когорта (17 0/7- 28 6/7 недель)
Валидированный интервал (191/7-206/7)
Переменные
Случаи
N (%)
(N=217)
Контроли
N (%)
(N=4,292)
P-значение
Случаи
N (%)
(N=81)
Контроли
N (%)
(N=162)
P-значение
Случаи
N (%)
(N=18)
Контроли
N (%)
(N=36)
P-значение
Материнские характеристики
Возраст матери при включении в исследование, годы
0,245
0,239
0,387
18-22 лет
58 (26,7)
990 (23,1)
22 (27,2)
47 (29,0)
6 (33,3)
13 (36,1)
23-27 лет
56 (25,8)
1,222 (28,5)
17 (21,0)
41 (25,3)
6 (33,3)
9 (25,0)
28-32 лет
54 (24,9)
1,154 (26,9)
25 (30,9)
34 (21,0)
5 (27,8)
5 (13,9)
33-37 лет
31 (14,3)
692 (16,1)
9 (11,1)
30 (18,5)
1 (5,6)
7 (19,4)
38 или больше
18 (8,3)
234 (5,5)
8 (9,9)
10 (6,2)
0
2 (5,6)
Среднее
28
28
28
28
25
27
Медиана
27
27
28
27
25
25
Интерквартильный размах
22-32
23-32
21-32
22-32
21-30
22-33
Индекс массы тела, кг/м2
0,380
0,802
0,959
Меньше 18,5
10 (4,7)
129 (3,1)
1 (1,3)
2 (1,3)
0
0
18,5-24,9
78 (36,8)
1,789 (42,3)
25 (31,3)
55 (34,6)
8 (44,4)
16 (45,7)
25,0-29,9
54 (25,5)
1,091 (25,8)
26 (32,5)
46 (28,9)
4 (22,2)
9 (25,7)
30,0-34,9
39 (18,4)
617 (15,6)
17 (21,3)
25 (15,7)
3 (16,7)
4 (11,4)
35,0-39,9
17 (8,0)
320 (7,6)
6 (7,5)
17 (10,7)
2 (11,1)
5 (14,3)
Больше 40,0
14 (6,6)
286 (6,7)
5 (6,3)
14 (8,8)
1 (5,6)
1 (2,9)
Среднее
27,8
27,5
28,4
29,1
28,2
27,4
Медиана
26,5
25,7
27,4
27,8
26,5
27
Интерквартильный размах
22,7-31,8
22,3-31,1
23,6-32,0
23,4-32,4
23,8-33,7
22,3-30,6
Уровень образования
<0,0002
0,201
0,263
Ученая степень
13 (6,0)
461 (10,9)
6 (7,7)
14 (8,7)
0
2 (5,7)
Диплом университета
34 (15,8)
701 (16,6)
10 (12,6)
22 (13,8)
2 (11,1)
5 (14,3)
Любой колледж
51 (23,7)
936 (22,2)
19 (24,0)
23 (14,4)
1 (5,6)
5 (14,3)
Аттестат зрелости/эквивалент
46 (21,4)
1,032 (24,5)
16 (20,2)
50 (31,3)
5 (27,8)
14 (40,0)
Любая средняя школа
53 (24,6)
774 (18,4)
25 (31,6)
36 (22,5)
9 (50,0)
6 (17,1)
9 классов или меньше
12 (5,8)
292 (6,9)
3 (3,8)
14 (8,7)
1 (5,6)
3 (8,6)
Другое
6 (2,8)
23 (0,6)
0
1 (0,6)
0
0
Этническая принадлежность
0,157
0,035
0,844
Испанцы или латино
89 (41,0)
1,557 (36,3)
27 (33,3)
77 (47,5)
7 (38,9)
15 (41,7)
Не испанцы или латино
128 (59,0)
2,735 (63,7)
54 (66,7)
85 (52,5)
11 (61,1)
21 (58,3)
Раса
0,887
0,811
0,319
Американские индейцы/коренные жители Аляски
1 (0,5)
29 (0,7)
0
2 (1,2)
0
1 (2,8)
Азиатская
4 (1,8)
131 (3,1)
1 (1,2)
1 (0,6)
0
1 (2,8)
Черная или афро-американцы
45 (20,7)
838 (19,5)
19 (23,5)
37 (22,8)
2 (11,1)
11 (30,6)
Коренные гавайцы или другие обитатели тихоокеанских островов
0
12 (0,30)
0
2 (1,2)
0
1 (2,8)
Белая
156 (71,9)
3,101 (72,3)
58 (71,6)
114 (70,4)
16 (88,9)
22 (61,1)
Другое
11 (5,1)
193 (4,5)
3 (3,7)
6 (3,7)
0
0
Акушерские характеристики
Первая беременность
64 (29,5)
1,212 (28,2)
0,689
27 (33,3)
39 (24,1)
0,126
5 (27,8)
8 (22,2)
0,652
Повторная беременность
153 (70,5)
3,080 (71,8)
54 (66,7)
123 (75,9)
13 (72,2)
28 (77,8)
Число предшествующих полносрочных родоразрешений
0,007
0,326
0,790
1 или больше
113 (73,8)
2,538 (82,4)
40 (74,5)
102 (82,9)
10 (76,9)
22 (78,6)
Нет
40 (26,2)
542 (17,6)
13 (24,5)
21 (17,1)
3 (23,1)
6 (21,4)
Число предшествующих самопроизвольных PTD
<0,0001
0,221
0,524
1 или больше
35 (22,9)
339 (11,0)
9 (16,7)
11 (8,9)
1 (7,7)
6 (21,4)
Нет
118 (77,1)
2,741 (89,0)
45 (83,3)
112 (91,1)
12 (92,3)
22 (78,6)
Характеристики образа жизни
Курение
0,412
0,719
1,000
Да
34 (15,7)
588 (13,7)
15 (18,5)
27 (16,7)
3 (16,7)
6 (16,7)
Нет
183 (84,3)
3,704 (86,3)
66 (81,5)
135 (83,3)
15 (83,3)
30 (83,3)
Запрещенные средства
0,283
0,628
0,739
Да
16 (7,4)
242 (5,6)
6 (7,4)
15 (9,3)
2 (11,1)
3 (8,3)
Нет
201 (92,6)
4,050 (94,4)
75 (92,6)
147 (90,7)
16 (88,9)
33 (91,7)
Алкоголь
0,096
0,628
0,278
Да
20 (9,2)
273 (6,4)
6 (7,4)
15 (9,3)
4 (22,2)
4 (11,1)
Нет
197 (90,8)
4,018 (93,6)
75 (92,6)
147 (90,7)
14 (77,8)
32 (88,9)
Употребление алкоголя
0,108
0,592
0,278
Да (количество не известно)
3 (1,4)
39 (0,9)
0
2 (1,2)
0
0
Социально (нерегулярное)
16 (7,4)
230 (5,4)
6 (7,4)
13 (8,0)
4 (22,2)
4 (11,1)
Тяжелое (ежедневное)
1 (0,5)
4 (0,09)
0
0
0
0
Нет
197 (90,8)
4,018 (93,6)
75 (92,6)
147 (90,7)
14 (77,8)
32 (88,9)
Медицинские характеристики
Кровотечение во время беременности после 12 нед.
0,006
0,360
0,308
Да
21 (9,7)
228 (5,3)
7 (8,6)
9 (5,6)
0
2 (5,6)
Нет
196 (90,3)
4,064 (94,7)
74 (91,4)
153 (94,4)
18 (100,0)
34 (94,4)
sPTD, самопроизвольное преждевременное родоразрешение; PTD, преждевременное родоразрешение; N, число субъектов.
Сравнения клинических данны4 между случаями и контролями осуществляли с использованием критерия хи-квадрат или точного критерия Фишера или критерия Манна-Уитни,в зависимости от ситуации (система SAS 9.4) и R (3.1.0).
Отсутствующие значения исключены из частотных таблиц.
Таблица 4
Исследование
Верификация
Валидация
№ образца (17-28 нед.)*
86, 172
50, 100
81, 162
№ образца (все BMI)*
22, 44†
9, 18†
18, 36‡
AUC (все BMI)
0,74 (p=8e-4)†
0,77 (p=0,01)†
0,67 (p=0,02)‡
№ образца (BMI <35)*
17, 33†
6, 17†
15, 29‡
AUC (BMI <35)
0,79 (p=3e-4)†
0,79 (p=0,015)†
0,70 (p=0,02)‡
Эквивалентность теста p-значений и AUC=0,5 с помощью односторонней статистики Уилкоксона-Манна-Уитни
* Число случаев, число контролей
†GA при взятии крови, недели 19/0-21/6
‡Оптимальный интервал GA при взятии крови из валидации фиксированной последовательности (19/1-20/6)
Таблица 5
BMI (кг/м2)
AUROC
Все BMI
0,67 (p=0,044)
BMI меньше или равен 45
0,67 (p=0,047)
BMI меньше или равен 40
0,68 (p=0,048)
BMI меньше или равен 37
0,71 (p=0,020)
BMI больше 18
0,67 (p=0,047)
BMI больше 20
0,65 (p=0,087)
BMI больше 22
0,69 (p=0,048)
BMI больше 22 и меньше или равен 37
0,75 (p=0,016)
p-значения, определяемые с помощью статистики Уилкоксона-Манна-Уитни
GA при взятии крови, недели 19/1-20/6
Таблица 6: RBM скрининг
Ранний интервал (17-22 недели)
Средний интервал (23-25 недели)
Поздний интервал (26-28 недели)
АНАЛИЗИРУЕМОЕ ВЕЩЕСТВО
AUC
АНАЛИЗИРУЕМОЕ ВЕЩЕСТВО
AUC
АНАЛИЗИРУЕМОЕ ВЕЩЕСТВО
AUC
Фибриноген
0,76
Васкулярная молекула клеточной адгезии 1, VCAM 1
0,96
Аполипопротеин C III, Apo C III
0,97
Антилейкопротеиназа, ALP
0,75
Рецептор эпидермального фактора роста, EGFR
0,79
Аполипопротеин B, Apo B
0,85
Молекула повреждения почки 1, KIM 1
0,73
Молекула клеточной адгезии, родственная эмбриональному опухолевому антигену 1, CEACAM1
0,78
Аполипопротеин E, Apo E
0,85
Тканевой ингибитор металлопротеиназы 1, TIMP 1
0,72
Молекула клеточной адгезии, родственная эмбриональному опухолевому антигену 6, CEACAM6
0,76
Глутатион-S-трансфераза α, GSTα
0,82
β2 микроглобулин, B2M
0,69
Ангиотензиноген
0,75
Связывающий инсулиноподобный фактор роста белок 6, IGFBP6
0,81
Фактор трилистника 3, TFF3
0,69
β-субъединица рецептора интерлейкина 6, IL 6R β
0,75
Тирозинкиназа с Ig и EGF гомологичными доменами 1, Tie 1
0,78
Тирозинкиназа с Ig и EGF гомологичными доменами 2, TIE 2
0,69
CD5-подобный антиген, CD5L
0,72
Связывающий инсулиноподобный фактор роста белок 4, IGFBP4
0,78
Ангиотензиноген
0,67
Легочный сурфактант-ассоциированный белок D, SP D
0,7
Фактор стволовых клеток, SCF
0,77
P селектин
0,67
Катепсин B pro CTSB
0,7
Фосфосеринаминотрансфераза, PSAT
0,76
Пепсиноген I, PGI
0,66
Гормон роста, GH
0,69
Фактор трилистника 3, TFF3
0,72
Специфичный антиген предстательной железы, общий, tPSA
0,66
β-микросеминопротеин, PSP94
0,69
Хемокин CC 4, HCC 4
0,71
Легочный хемокин, регулируемый активацией, PARC
0,66
Серотрансферрин, трансферрин
0,68
Макрофагальный колониестимулирующий фактор 1, M CSF
0,7
Амилоидный P компонент сыворотки, SAP
0,66
Молекула адгезии нервных клеток, Nr CAM
0,67
Родственный фактору H комплемента белок 1, CFHR1
0,7
Рецептор фактора некроза опухоли I, TNF RI
0,66
Активатор плазминогена урокиназного типа, uPA
0,67
Тирозинкиназа с Ig и EGF гомологичными доменами 2, TIE 2
0,69
Рецептор фактора роста эндотелия сосудов 3, VEGFR 3
0,66
Витронектин
0,65
Специфичный антиген предстательной железы общий, tPSA
0,68
Катепсин D
0,65
Фактор фон Виллебранда, vWF
0,65
Глюкагоноподобный пептид 1 общий, GLP 1 общий
0,68
Фетуин A
0,65
Тестостерон общий
0,65
Рецептор FASLG, FAS
0,67
Молекула адгезии тромбоцит-эндотелиальных клеток, PECAM 1
0,65
Липокалин 1, LCN1
0,65
Связывающий инсулиноподобный фактор роста белок 5, IGFBP5
0,67
Кадгерин 1, E Cad
0,64
Антиген плоскоклеточной карциномы 1, SCCA 1
0,64
Рецептор активатора плазминогена урокиназного типа, uPAR
0,66
Антиген злокачественной опухоли 15.3, CA 15.3
0,64
Моноцитарный хемотаксический белок 1, MCP 1
0,64
Гаптоглобин
0,66
Прогестерон
0,64
Тенасцин C, TN C
0,63
Связывающий инсулиноподобный фактор роста белок 2, IGFBP 2
0,66
Тенасцин C, TN C
0,64
Комплемент C3, C3
0,63
Калликреин 5
0,66
Альдозоредуктаза
0,63
Гликопротеин мочи Тамма-Хорсфалла, THP
0,63
Тенасцин C, TN C
0,65
Ангиопоэтин 1, ANG 1
0,63
Нейропилин 1
0,63
Катепсин D
0,65
Аполипопротеин A II, Apo A II
0,63
Мидкин
0,62
Рецептор фактора некроза опухоли I, TNF RI
0,65
Остеопротегерин, OPG
0,63
Кортизол, Кортизол
0,62
Антагонист рецептора интерлейкина 1, IL 1ra
0,64
Фолликулостимулирующий гормон, FSH
0,62
Иммуноглобулин M, IgM
0,62
Проинсулин интактный
0,64
Онкоген альфа, связанный с ростом, GRO α
0,62
Рецептор для конечных продуктов усиленного гликозилирования, RAGE
0,62
Проинсулин общий
0,64
Матриксная металлопротеиназа 7, MMP 7
0,62
N-концевой прогормон натрийуретического пептида головного мозга, NT proBNP
0,62
Матриксная металлопротеиназа 9, MMP 9
0,64
Фосфосеринаминотрансфераза, PSAT
0,62
Макрофагальный белок воспаления 3β, MIP 3β
0,62
Ингибиторный фактор миелоидны клеток-предшественников 1, MPIF 1
0,64
Серотрансферрин, трансферрин
0,62
Молекула повреждения почки 1, KIM 1
0,61
Ангиопоэтин 2, ANG 2
0,64
Аполипопротеин C III, Apo C III
0,61
Матриксная металлопротеиназа 9, общая, MMP 9 общая
0,61
Фактор фон Виллебранда, vWF
0,63
Карбоангидраза 9, CA 9
0,61
Клеточный фибронектин, cFib
0,6
Цистатин B
0,63
Комплемент C3, C3
0,61
Мезотелин, MSLN
0,6
Комплемент C3, C3
0,62
Цистатин C
0,61
Транстиретин TTR
0,6
Мидкин
0,62
Связывающий инсулиноподобный фактор роста белок 4, IGFBP4
0,61
Коллаген IV
0,6
Матриксная металлопротеиназа 9 общая, MMP 9 общая
0,62
Молекула межклеточной адгезии 1, ICAM 1
0,61
Монокин, индуцируемый интерфероном γ, MIG
0,62
Макрофагальный колониестимулирующий фактор 1, M-CSF
0,61
Легочный хемокин, регулируемый активацией, PARC
0,62
Мидкин
0,61
α-рецептор интерлейкина 2, α-рецептор IL 2
0,62
Ангиогенин
0,6
β-субъединица рецептора интерлейкина 6, IL 6R β
0,61
C-реактивный белок, CRP
0,6
CD5-подобный антиген, CD5L
0,61
Антиген CD 40, CD40
0,6
Гепсин
0,61
Клеточный фибронектин, cFib
0,6
Фетуин A
0,61
α-рецептор интерлейкина 2, α-рецептор IL 2
0,6
Хемоаттрактант B-лимфоцитов, BLC
0,61
Тромбоспондин 4, TSP4
0,6
Антилейкопротеиназа, ALP
0,61
α2 макроглобулин, A2Macro
0,61
Тканевой ингибитор металлопротеиназы 1 TIMP 1
0,61
Рецептор для конечных продуктов усиленного гликозилирования, RAGE
0,6
Панкреатический секреторный ингибитор трипсина, TATI
0,6
Адипонектин
0,6
Люмикан
0,6
Аполипопротеин C I, Apo C I
0,6
Аполипопротеин H, Apo H
0,6
Фактор роста гепатоцитов, HGF
0,6
Таблица 7: ранжирование анализируемых веществ в раннем интервале (GABD 17-22 нед.) с помощью различных многомерных моделей
Ранг
rf
бустинг
лассо
логистический
1
Фактор активации B-клеток, BAFF
Макрофагальный белок воспаления 3β, MIP 3β
Антилейкопротеиназа, ALP
Аполипопротеин A II, Apo A II
2
Макрофагальный белок воспаления 3β, MIP 3β
Фактор активации B-клеток, BAFF
Ангиотензиноген
Аполипопротеин a, Lp a
3
Фибриноген
Молекула повреждения почек 1, KIM 1
Молекула повреждения почек 1 KIM 1
Аполипопротеин A IV, Apo A IV
4
Молекула повреждения почек 1, KIM 1
Фибриноген
Прогестерон
Аполипопротеин B, Apo B
5
Тканевой ингибитор металлопротеиназ 1, TIMP 1
β2 микроглобулин, B2M
Моноцитарный хемотаксический белок 1 MCP 1
Аполипопротеин C III, Apo C III
6
Член суперсемейства лигандов фактора некроза опухоли 12, Tweak
Тканевой ингибитор металлопротеиназ 1, TIMP 1
Фолликулостимулирующий гормон, FSH
Аполипопротеин H, Apo H
7
Легочный хемокин, регулируемый активацией, PARC
N-концевой прогормон натрийуретического пептида головного мозга, NT proBNP
Серотрансферрин, трансферрин
Ангиотензинпревращающий фермент, ACE
8
Панкреатический полипептид, PPP
Антилейкопротеиназа, ALP
Тирозинкиназа с Ig и EGF гомологичными доменами, 2 TIE 2
Аполипопротеин C I, Apo C I
9
Ангиотензиноген
Специфичный антиген предстательной железы, общий, tPSA
Тироглобулин, TG
Хемоаттрактант B-лимфоцитов, BLC
10
Специфичный антиген предстательной железы, общий, tPSA
Панкреатический полипептид, PPP
Антиген злокачественной опухоли 15.3, CA 15.3
Адипонектин
11
Антилейкопротеиназа, ALP
Коллаген IV
Легочный хемокин, регулируемый активацией, PARC
Ангиотензиноген
12
Тирозинкиназа с Ig и EGF гомологичными доменами 2, TIE 2
Тирозинкиназа с Ig и EGF гомологичными доменами 2, TIE 2
Фактор трилистника 3, TFF3
AXL рецепторная тирозинкиназа, AXL
13
Катепсин D
Ангиотензиноген
Мидкин
Ангиогенин
14
β2 микроглобулин, B2M
Катепсин D
β2 микроглобулин, B2M
Альдозоредуктаза
Таблица 8: ранжирование анализируемых веществ в среднем интервале (GABD 23-25 нед.) с помощью различных многомерных моделей
Ранг
rf
бустинг
лассо
логистический
1
Васкулярная молекула клеточной адгезии 1, VCAM 1
Васкулярная молекула клеточной адгезии 1, VCAM 1
Васкулярная молекула клеточной адгезии 1, VCAM 1
α-фетопротеин, AFP
2
Альдозоредуктаза
Альдозоредуктаза
Остеопротегерин, OPG
Адипонектин
3
Остеопротегерин, OPG
Остеопротегерин, OPG
Аполипопротеин E, Apo E
Ангиогенин
4
Связывающий инсулиноподобный фактор роста белок 3, IGFBP 3
Ангиотензиноген
Альдозоредуктаза
α1 антитрипсин, AAT
5
Молекула клеточной адгезии, родственная эмбриональному опухолевому антигену 1, CEACAM1
Аполипопротеин E, Apo E
Антиген злокачественной опухоли 72.4, CA 72.4
Ангиотензинпревращающий фермент, ACE
6
Ангиотензиноген
Интерлейкин 16, IL 16
Рецептор эпидермального фактора роста, EGFR
α1 микроглобулин, A1Micro
7
Интерлейкин 16, IL 16
Рецептор эпидермального фактора роста, EGFR
Прогестерон
α1 антихимотрипсин, AACT
8
Связывающий инсулиноподобный фактор роста белок 4, IGFBP4
α-фетопротеин, AFP
Хорионический гонадотропин β человека, hCG
α2 макроглобулин, A2Macro
9
Рецептор эпидермального фактора роста, EGFR
α2 макроглобулин, A2Macro
Тканевой ингибитор металлопротеиназ 1, TIMP 1
Альдозоредуктаза
10
Молекула клеточной адгезии, родственная эмбриональному опухолевому антигену 6, CEACAM6
Ангиопоэтин 2, ANG 2
α-фетопротеин, AFP
X6Ckine
11
Активатор плазминогена урокиназного типа, uPA
Аполипопротеин A, Lp a
Молекула клеточной адгезии, родственная эмбриональному опухолевому антигену 1, CEACAM1
Ангиопоэтин 2, ANG 2
12
Тканевой ингибитор металлопротеиназ 1, TIMP 1
α1 микроглобулин, A1Micro
Связывающий инсулиноподобный фактор роста белок 5, IGFBP5
Белок A, родственный цепи MHC класса I, MICA
13
Прогестерон
Ангиогенин
Лектинподобный рецептор окисленных LDL 1, LOX 1
Ассоциированный с желатиназой нейтрофилов липокалин, NGAL
14
β-субъединица рецептора интерлейкина 6, IL 6R β
Фактор активации B-клеток, BAFF
Рецептор FASLG, FAS
P селектин
15
Тромбоспондин 4, TSP4
Адипонектин
Серотрансферрин, трансферрин
Тканевой ингибитор металлопротеиназ 2, TIMP 2
Таблица 9: ранжирование анализируемых веществ в позднем интервале (GABD 26-28 недели) с помощью различных многомерных моделей
Ранг
rf
бустинг
лассо
логистический
1
Аполипопротеин C III, Apo C III
Аполипопротеин C III, Apo C III
Аполипопротеин C III, Apo C III
α1 микроглобулин, A1Micro
2
Аполипопротеин E, Apo E
Аполипопротеин E, Apo E
Интерлейкин 18, IL 18
Альдозоредуктаза
3
Связывающий инсулиноподобный фактор роста белок 4, IGFBP4
EN RAGE
Аполипопротеин B, Apo B
Ангиогенин
4
Связывающий инсулиноподобный фактор роста белок 6, IGFBP6
α-фетопротеин, AFP
Глутатион-S-трансфераза α, GST α
α-фетопротеин, AFP
5
Аполипопротеин B, Apo B
Аполипопротеин B, Apo B
Тирозинкиназа с Ig и EGF гомологичными доменами 1, Tie 1
α1 антихимотрипсин, AACT
6
Интерлейкин 18, IL 18
Ангиотензиноген
Фактор трилистника 3, TFF3
α1 антитрипсин, AAT
7
Глутатион-S-трансфераза α, GST α
Ангиотензинпревращающий фермент, ACE
Аполипопротеин E, Apo E
X6Ckine
8
Фактор стволовых клеток, SCF
Альдозоредуктаза
Связывающий инсулиноподобный фактор роста белок 4, IGFBP4
α2 макроглобулин, A2Macro
9
Фосфосеринаминотрансфераза, PSAT
Аполипопротеин A, Lp a
Рецептор FASLG, FAS
Адипонектин
10
Тирозинкиназа с Ig и EGF гомологичными доменами 1, Tie 1
Ангиопоэтин 2, ANG 2
Тирозинкиназа с Ig и EGF гомологичными доменами 2, TIE 2
Хромогранин A, CgA
11
EN RAGE
X6Ckine
Креатинкиназа MB, CK MB
Макрофагальный белок воспаления 1β, MIP 1β
12
Рецептор фактора роста эндотелия сосудов 1, VEGFR 1
α2 макроглобулин, A2Macro
Фактор стволовых клеток, SCF
Ангиопоэтин 2, ANG 2
13
Связывающий инсулиноподобный фактор роста белок 2, IGFBP 2
Ангиогенин
Хемокин CC 4, HCC 4
Ангиотензинпревращающий фермент, ACE
14
Хемокин CC 4, HCC 4
α1 антихимотрипсин, AACT
Родственный фактору H комплемента белок 1, CFHR1
Ангиотензиноген
15
Тирозинкиназа с Ig и EGF гомологичными доменами 2, TIE 2
Адипонектин
α2 макроглобулин, A2Macro
Аполипопротеин A, Lp a
Таблица 10: таблица с информацией о эпитопах и клональности для комплектов, тестированных по анализируемым веществам IBP4_HUMAN И SHBG_HUMAN, когда доступно.
Анализируемое вещество
Продавец
Номер по каталогу
Иммобилизованное антитело
Идентифицирующее антитело
Эпитопы
Значение r корреляции Пирсона, ELISA в сравнении с MS
IBP4_HUMAN
ANSCH Labs
Webster, Texas
AL-126
Моноклональное
Моноклональное
C-концевые
0,8631
SHBG_HUMAN
R&D Systems
Minneapolis, Minnesota
DSHBG0B
Моноклональное
Моноклональное
Не картированы
0,9228
SHBG_HUMAN
Raybiotech
Norcross, Georgia
ELH-SHBG
Моноклональное
Моноклональное
Не картированы
0,8675
IBP4_HUMAN
ABNOVA
Taipei, Taiwan
KA1873
Моноклональное
Моноклональное
Не известно
0,2635
IBP4_HUMAN
ABCAM
Cambridge, Massachusetts
ab100542
Моноклональное
Моноклональное
Asp22-Glu258
0,3439
IBP4_HUMAN
ANSCH Labs
Webster, Texas
AL-128
Моноклональное
Моноклональное
N-конец и C-конец
0,2954
Таблица 11: анализируемые вещества, показывающие комплекты, которые или коррелируют с MS данными или нет.
Коррелирующие данные
Не коррелирующие данные
Анализируемое вещество (№ комплекта ELISA)
Значение r корреляции Пирсона, ELISA в сравнении с MS
Анализируемое вещество (№ комплекта ELISA)
Значение r корреляции Пирсона, ELISA в сравнении с MS
ANGT_HUMAN #1
0,6192
A2GL_HUMAN
-0,2933
B2MG_HUMAN
0,8414
ANGT_HUMAN #2
-0,01351
BGH3_HUMAN
0,7159
APOH_HUMAN
0,2669
C06_HUMAN
0,8045
CHL1_HUMAN
0,0795
CD14_HUMAN
0,8004
CLUS_HUMAN
0,3132
CHL1_HUMAN
0,9271
CPN1_HUMAN
0,1775
FETUA_HUMAN
0,7259
CSH_HUMAN
-0,3172
IBP4_HUMAN#1
0,8631
FBLN1_HUMAN
0,1141
IGF2_HUMAN
0,6346
IBP4_HUMAN #2
0,3439
LBP_HUMAN
0,7389
IBP4_HUMAN #3
0,2365
PAPP1_HUMAN #1
0,9163
IBP4_HUMAN #4
0,2954
SHBG_HUMAN #1
0,9228
PAPP1_HUMAN #2
0,04381
SHBG_HUMAN #2
0,8675
PRG2_HUMAN #1
0,2699
PSG2_HUMAN #2
-0,06944
PTGDS_HUMAN
0,1627
TENX_HUMAN
-0,1116
TIE1_HUMAN
0,0384
VTDB_HUMAN
-0,2459
VTNC_HUMAN
0,1243
Таблица 12: комплекты ELISA для IBP4 и SHBG, демонстрирующие разделение (одномерное) на sPTB и контроль
Анализируемое вещество
R Пирсона
P-значение разделения на контроль и случай
Продавец
№ по каталогу
IBP4_HUMAN
0,8631
0,0009
ANSCH Labs
Webster, Texas
AL-126
SHBG_HUMAN
0,8675
0,0374
R&D Systems
Minneapolis, Minnesota
DSHBG0B
Таблица 13
dbSNP rs# id кластера
Гетеро-зиготность
Валидация
MAF
Функция
Аллель dbSNP
Остаток белка
Положение кодона
Аминокислотн. полож. в NP_001543.2
мРНК полож. в NM_001552.2
rs757185079
0
миссенс
C
Pro [P]
2
214
953
эталонный контиг
A
Gln [Q]
2
214
rs759609271
0
нонсенс
T
1
222
976
эталонный контиг
C
Gln [Q]
1
222
rs765360682
0
миссенс
A
His [H]
2
223
980
эталонный контиг
G
Arg [R]
2
223
Таблица 14
dbSNP rs# id кластера
Гетеро-зиготность
Валидация
MAF
Функция
Аллель dbSNP
Остаток белка
Положение кодона
Аминокислотн. полож. в NP_001031.2
мРНК полож. в NM_001040.2
rs751519873
0
миссенс
T
Val [V]
2
171
591
эталонный контиг
C
Ala [A]
2
171
rs528701583
0
по кластеру
Синоним
A
Ala [A]
3
171
592
эталонный контиг
G
Ala [A]
3
171
rs201120578
0
по кластеру С использованием 1000GenomeData
0,0002
Миссенс
T
Phe [F]
1
172
593
эталонный контиг
C
Leu [L]
1
172
rs747379879
0
Синоним
C
Leu [L]
3
172
595
эталонный контиг
T
Leu [L]
3
172
rs769030967
0
Миссенс
C
Arg [R]
1
173
596
эталонный контиг
G
Gly [G]
1
173
rs777068397
0
Миссенс
C
Ala [A]
2
173
597
эталонный контиг
G
Gly [G]
2
173
rs367555757
0
по кластеру
Синоним
A
Gly [G]
3
173
598
Синоним
C
Gly [G]
3
173
эталонный контиг
G
Gly [G]
3
173
rs567677603
0
по кластеру
Синоним
T
Pro [P]
3
178
613
эталонный контиг
C
Pro [P]
3
178
rs115336700
0,01
по кластеру по частоте с использованием 1000GenomeData
0,0048
миссенс
C
Pro [P]
1
179
614
эталонный контиг
G
Ala [A]
1
179
rs765896254
0
миссенс
G
Ser [S]
2
181
621
эталонный контиг
A
Asn [N]
2
181
rs143134553
0
по кластеру с использованием 1000GenomeData
0,0002
миссенс
A
Lys [K]
3
181
622
эталонный контиг
C
Asn [N]
3
181
rs139379650
0
миссенс
T
Trp [W]
1
183
626
эталонный контиг
C
Arg [R]
1
183
rs759318203
0
миссенс
A
Gln [Q]
2
183
627
эталонный контиг
G
Arg [R]
2
183
Таблица 15
Исследование_BMI>22≤37
Исследование_все_BMI
Исследование_BMI<35
GABD (сутки)
AUC
MW p-значение
Случаи
Контроль
AUC
MW p-значение
Случаи
Контроль
AUC
MW p-значение
Случаи
Контроль
133:143
0,822
0,0018
10
18
0,719
0,0064
16
32
0,788
0,0023
12
22
133:144
0,791
0,0031
11
20
0,702
0,0089
17
34
0,763
0,0037
13
24
133:145
0,786
0,0027
12
21
0,711
0,0053
18
36
0,766
0,0023
14
26
133:146
0,788
0,0023
12
22
0,726
0,0025
19
38
0,773
0,0016
14
28
133:147
0,804
0,001
13
22
0,730
0,0016
20
40
0,791
0,0006
15
29
133:148
0,804
0,001
13
22
0,730
0,0016
20
40
0,791
0,0006
15
29
133:149
0,804
0,001
13
22
0,730
0,0016
20
40
0,791
0,0006
15
29
133:150
0,804
0,001
13
22
0,730
0,0016
20
40
0,791
0,0006
15
29
133:151
0,773
0,0023
14
23
0,722
0,0018
21
42
0,778
0,0007
16
31
133:152
0,773
0,0023
14
23
0,722
0,0018
21
42
0,778
0,0007
16
31
133:153
0,787
0,0009
15
25
0,736
0,0008
22
44
0,790
0,0003
17
33
Верификация_BMI>22≤37
Верификация_все_BMI
Верификация_BMI<35
GABD (сутки)
AUC
MW p-значение
Случаи
Контроль
AUC
MW p-значение
Случаи
Контроль
AUC
MW p-значение
Случаи
Контроль
133:143
0,889
0,0364
2
9
0,750
0,0415
6
12
0,818
0,0278
4
11
133:144
0,889
0,0364
2
9
0,750
0,0415
6
12
0,818
0,0278
4
11
133:145
0,867
0,0245
3
10
0,765
0,023
7
14
0,815
0,0175
5
13
133:146
0,867
0,0245
3
10
0,765
0,023
7
14
0,815
0,0175
5
13
133:147
0,867
0,0245
3
10
0,765
0,023
7
14
0,815
0,0175
5
13
133:148
0,813
0,0291
4
12
0,727
0,0351
8
16
0,767
0,0274
6
15
133:149
0,839
0,0173
4
14
0,772
0,01
9
18
0,794
0,0149
6
17
133:150
0,839
0,0173
4
14
0,772
0,01
9
18
0,794
0,0149
6
17
133:151
0,839
0,0173
4
14
0,772
0,01
9
18
0,794
0,0149
6
17
133:152
0,839
0,0173
4
14
0,772
0,01
9
18
0,794
0,0149
6
17
133:153
0,839
0,0173
4
14
0,772
0,01
9
18
0,794
0,0149
6
17
Валидация_BMI>22≤37
Валидация_все_BMI
Валидация_BMI<35
GABD (сутки)
AUC
MW p-значение
Случаи
Контроль
AUC
MW p-значение
Случаи
Контроль
AUC
MW p-значение
Случаи
Контроль
133-143
0,867
0,0051
7
15
0,698
0,0572
12
24
0,766
0,0248
9
19
133-144
0,768
0,0185
10
19
0,670
0,058
16
32
0,695
0,0501
13
26
133-145
0,788
0,0073
11
21
0,685
0,0324
17
34
0,707
0,0305
14
28
133-146
0,750
0,0157
12
23
0,670
0,0438
18
36
0,697
0,0342
15
29
133-147
0,750
0,0157
12
23
0,670
0,0438
18
36
0,697
0,0342
15
29
133-148
0,750
0,0157
12
23
0,670
0,0438
18
36
0,697
0,0342
15
29
133-149
0,684
0,0587
14
26
0,623
0,127
20
40
0,649
0,091
17
32
133-150
0,684
0,0587
14
26
0,623
0,127
20
40
0,649
0,091
17
32
133-151
0,609
0,2457
16
27
0,555
0,4782
22
43
0,598
0,2489
19
33
133-152
0,628
0,1582
17
28
0,573
0,3327
23
46
0,609
0,1897
20
34
133-153
0,646
0,0988
18
29
0,574
0,3152
24
48
0,609
0,1897
20
34
GABD (сутки) относится к интервалу гестационного возраста при взятии крови в сутках
Эквивалентность теста MW p-значений и AUC=0,5 с помощью односторонней статистики Уилкоксона-Манна-Уитни
Таблица 16: Сводка площадей пиков для переходов для четырех синтетических тяжелых пептидов IBP4_HUMAN
Последовательность пептида
SEQ ID №
Название белка
Mz предшественника
Заряд предшественника
Mz продукта
Заряд продукта
Фрагментарный ион
Время удержания
Площадь
LPGGLEPK
1
IBP4_HUMAN
409,75
2
211,14
1
b2
4,62
252768
LPGGLEPK
1
IBP4_HUMAN
409,75
2
325,19
1
b4
4,66
76266
LPGGLEPK
1
IBP4_HUMAN
409,75
2
705,40
1
y7
4,66
844128
LPGGLEPK
1
IBP4_HUMAN
409,75
2
608,35
1
y6
4,66
866360
LPGGLEPK
1
IBP4_HUMAN
409,75
2
551,33
1
y5
4,62
96412
LPGGLEPK
1
IBP4_HUMAN
409,75
2
252,18
1
y2
4,66
939572
LPGGLEPK
1
IBP4_HUMAN
409,75
2
353,20
2
y7
4,66
3489414
QCHPALDGQR
2
IBP4_HUMAN
596,28
2
289,10
1
b2
1,85
9703
QCHPALDGQR
2
IBP4_HUMAN
596,28
2
426,16
1
b3
1,85
14713
QCHPALDGQR
2
IBP4_HUMAN
596,28
2
822,36
1
b7
1,85
2136
QCHPALDGQR
2
IBP4_HUMAN
596,28
2
903,47
1
y8
1,85
17798
QCHPALDGQR
2
IBP4_HUMAN
596,28
2
766,41
1
y7
1,85
73221
QCHPALDGQR
2
IBP4_HUMAN
596,28
2
669,36
1
y6
1,85
5019
QCHPALDGQR
2
IBP4_HUMAN
596,28
2
598,32
1
y5
1,85
4078
QCHPALDGQR
2
IBP4_HUMAN
596,28
2
485,23
1
y4
1,85
4383
QCHPALDGQR
2
IBP4_HUMAN
596,28
2
370,21
1
y3
1,85
25859
QCHPALDGQR
2
IBP4_HUMAN
397,86
3
289,10
1
b2
1,85
57043
QCHPALDGQR
2
IBP4_HUMAN
397,86
3
426,16
1
b3
1,85
109467
QCHPALDGQR
2
IBP4_HUMAN
397,86
3
523,21
1
b4
1,85
12019
QCHPALDGQR
2
IBP4_HUMAN
397,86
3
594,25
1
b5
1,85
30122
QCHPALDGQR
2
IBP4_HUMAN
397,86
3
213,58
2
b3
1,85
7249
QCHPALDGQR
2
IBP4_HUMAN
397,86
3
262,11
2
b4
1,85
11989
QCHPALDGQR
2
IBP4_HUMAN
397,86
3
297,63
2
b5
1,85
71677
QCHPALDGQR
2
IBP4_HUMAN
397,86
3
354,17
2
b6
1,85
10532
QCHPALDGQR
2
IBP4_HUMAN
397,86
3
411,68
2
b7
1,85
8062
QCHPALDGQR
2
IBP4_HUMAN
397,86
3
766,41
1
y7
1,85
118740
QCHPALDGQR
2
IBP4_HUMAN
397,86
3
669,36
1
y6
1,85
79410
QCHPALDGQR
2
IBP4_HUMAN
397,86
3
598,32
1
y5
1,85
235615
QCHPALDGQR
2
IBP4_HUMAN
397,86
3
485,23
1
y4
1,85
637333
QCHPALDGQR
2
IBP4_HUMAN
397,86
3
370,21
1
y3
1,85
440794
QCHPALDGQR
2
IBP4_HUMAN
397,86
3
313,19
1
y2
1,85
26708
QCHPALDGQR
2
IBP4_HUMAN
397,86
3
532,25
2
y9
1,85
52271
QCHPALDGQR
2
IBP4_HUMAN
397,86
3
452,24
2
y8
1,85
24399
QCHPALDGQR
2
IBP4_HUMAN
397,86
3
383,71
2
y7
1,85
238180
QCHPALDGQR
2
IBP4_HUMAN
397,86
3
335,18
2
y6
1,85
64484
QCHPALDGQR
2
IBP4_HUMAN
397,86
3
299,66
2
y5
1,85
18478
QCHPALDGQR
2
IBP4_HUMAN
397,86
3
243,12
2
y4
1,85
50903
THEDLYIIPIPNCDR
3
IBP4_HUMAN
933,46
2
239,11
1
b2
8,25
35797
THEDLYIIPIPNCDR
3
IBP4_HUMAN
933,46
2
368,16
1
b3
8,25
15185
THEDLYIIPIPNCDR
3
IBP4_HUMAN
933,46
2
483,18
1
b4
8,25
14411
THEDLYIIPIPNCDR
3
IBP4_HUMAN
933,46
2
596,27
1
b5
8,25
19740
THEDLYIIPIPNCDR
3
IBP4_HUMAN
933,46
2
759,33
1
b6
8,25
35904
THEDLYIIPIPNCDR
3
IBP4_HUMAN
933,46
2
872,41
1
b7
8,25
38201
THEDLYIIPIPNCDR
3
IBP4_HUMAN
933,46
2
985,50
1
b8
8,25
38297
THEDLYIIPIPNCDR
3
IBP4_HUMAN
933,46
2
1195,64
1
b10
8,2
11054
THEDLYIIPIPNCDR
3
IBP4_HUMAN
933,46
2
1107,59
1
y9
8,25
22635
THEDLYIIPIPNCDR
3
IBP4_HUMAN
933,46
2
994,50
1
y8
8,25
33493
THEDLYIIPIPNCDR
3
IBP4_HUMAN
933,46
2
881,42
1
y7
8,25
98887
THEDLYIIPIPNCDR
3
IBP4_HUMAN
933,46
2
784,36
1
y6
8,25
2565
THEDLYIIPIPNCDR
3
IBP4_HUMAN
933,46
2
671,28
1
y5
8,25
31935
THEDLYIIPIPNCDR
3
IBP4_HUMAN
622,64
3
239,11
1
b2
8,25
13586
THEDLYIIPIPNCDR
3
IBP4_HUMAN
622,64
3
368,16
1
b3
8,25
6976
THEDLYIIPIPNCDR
3
IBP4_HUMAN
622,64
3
483,18
1
b4
8,25
12635
THEDLYIIPIPNCDR
3
IBP4_HUMAN
622,64
3
596,27
1
b5
8,25
36886
THEDLYIIPIPNCDR
3
IBP4_HUMAN
622,64
3
759,33
1
b6
8,25
138833
THEDLYIIPIPNCDR
3
IBP4_HUMAN
622,64
3
872,41
1
b7
8,25
190646
THEDLYIIPIPNCDR
3
IBP4_HUMAN
622,64
3
985,50
1
b8
8,25
54139
THEDLYIIPIPNCDR
3
IBP4_HUMAN
622,64
3
493,25
2
b8
8,25
13320
THEDLYIIPIPNCDR
3
IBP4_HUMAN
622,64
3
541,78
2
b9
8,25
6168
THEDLYIIPIPNCDR
3
IBP4_HUMAN
622,64
3
994,50
1
y8
8,25
5893
THEDLYIIPIPNCDR
3
IBP4_HUMAN
622,64
3
881,42
1
y7
8,25
297346
THEDLYIIPIPNCDR
3
IBP4_HUMAN
622,64
3
784,36
1
y6
8,25
14422
THEDLYIIPIPNCDR
3
IBP4_HUMAN
622,64
3
671,28
1
y5
8,25
212081
THEDLYIIPIPNCDR
3
IBP4_HUMAN
622,64
3
574,23
1
y4
8,25
10550
THEDLYIIPIPNCDR
3
IBP4_HUMAN
622,64
3
460,18
1
y3
8,25
4183
THEDLYIIPIPNCDR
3
IBP4_HUMAN
622,64
3
441,21
2
y7
8,25
59487
THEDLYIIPIPNCDR
3
IBP4_HUMAN
622,64
3
336,14
2
y5
8,25
48606
EDARPVPQGSCQSELHR
4
IBP4_HUMAN
659,31
3
245,08
1
b2
2,94
1864
EDARPVPQGSCQSELHR
4
IBP4_HUMAN
659,31
3
472,22
1
b4
2,94
7355
EDARPVPQGSCQSELHR
4
IBP4_HUMAN
659,31
3
569,27
1
b5
2,94
1648
EDARPVPQGSCQSELHR
4
IBP4_HUMAN
659,31
3
668,34
1
b6
2,94
30775
EDARPVPQGSCQSELHR
4
IBP4_HUMAN
659,31
3
765,39
1
b7
2,94
1033
EDARPVPQGSCQSELHR
4
IBP4_HUMAN
659,31
3
893,45
1
b8
2,94
6138
EDARPVPQGSCQSELHR
4
IBP4_HUMAN
659,31
3
950,47
1
b9
2,94
4242
EDARPVPQGSCQSELHR
4
IBP4_HUMAN
659,31
3
285,14
2
b5
2,94
680
EDARPVPQGSCQSELHR
4
IBP4_HUMAN
659,31
3
334,67
2
b6
2,94
1704
EDARPVPQGSCQSELHR
4
IBP4_HUMAN
659,31
3
383,20
2
b7
2,9
3561
EDARPVPQGSCQSELHR
4
IBP4_HUMAN
659,31
3
447,23
2
b8
2,94
3333
EDARPVPQGSCQSELHR
4
IBP4_HUMAN
659,31
3
475,74
2
b9
2,94
6911
EDARPVPQGSCQSELHR
4
IBP4_HUMAN
659,31
3
1083,49
1
y9
2,94
11083
EDARPVPQGSCQSELHR
4
IBP4_HUMAN
659,31
3
1026,47
1
y8
2,94
4195
EDARPVPQGSCQSELHR
4
IBP4_HUMAN
659,31
3
939,43
1
y7
2,94
8994
EDARPVPQGSCQSELHR
4
IBP4_HUMAN
659,31
3
779,40
1
y6
2,94
13897
EDARPVPQGSCQSELHR
4
IBP4_HUMAN
659,31
3
651,34
1
y5
2,94
23600
EDARPVPQGSCQSELHR
4
IBP4_HUMAN
659,31
3
564,31
1
y4
2,94
7164
EDARPVPQGSCQSELHR
4
IBP4_HUMAN
659,31
3
435,27
1
y3
2,94
8441
EDARPVPQGSCQSELHR
4
IBP4_HUMAN
659,31
3
322,19
1
y2
2,94
10343
EDARPVPQGSCQSELHR
4
IBP4_HUMAN
659,31
3
866,43
2
y15
2,94
2751
EDARPVPQGSCQSELHR
4
IBP4_HUMAN
659,31
3
830,91
2
y14
2,94
994
EDARPVPQGSCQSELHR
4
IBP4_HUMAN
659,31
3
752,86
2
y13
2,94
2065
EDARPVPQGSCQSELHR
4
IBP4_HUMAN
659,31
3
654,80
2
y11
2,94
26747
EDARPVPQGSCQSELHR
4
IBP4_HUMAN
659,31
3
542,25
2
y9
2,94
7872
Сравнительные MS данные для пептидов и переходов IBP4. Четыре различных сильно меченных пептида (R*+10 Да) являются примерами различных переходов их относительных интенсивностей, мониторинг которых можно осуществлять для количественного определения IBP4. Специалисты в данной области могут выбирать потенциально любые из этих пептидов или переходов или другие, которые не приведены в качестве примера, чтобы количественно определять IBP4.
Таблица 17: сводка площадей пиков для переходов для IBP4_HUMAN, измеряемого с использованием рекомбинантного белка
Последовательность пептида
SEQ ID №
Название белка
Mz предшественника
Заряд предшественника
Mz продукта
Заряд продукта
Фрагментарный ион
Время удержания
Площадь
CRPPVGCEELVR
5
sp|P22692|IBP4_HUMAN
491,24
3
317,14
1
b2
6,4
20339
CRPPVGCEELVR
5
sp|P22692|IBP4_HUMAN
491,24
3
414,19
1
b3
6,35
21220
CRPPVGCEELVR
5
sp|P22692|IBP4_HUMAN
491,24
3
610,31
1
b5
6,46
13132
CRPPVGCEELVR
5
sp|P22692|IBP4_HUMAN
491,24
3
667,33
1
b6
6,46
14894
CRPPVGCEELVR
5
sp|P22692|IBP4_HUMAN
491,24
3
207,60
2
b3
6,4
5605
CRPPVGCEELVR
5
sp|P22692|IBP4_HUMAN
491,24
3
256,13
2
b4
6,4
13853
CRPPVGCEELVR
5
sp|P22692|IBP4_HUMAN
491,24
3
414,19
2
b7
6,4
21460
CRPPVGCEELVR
5
sp|P22692|IBP4_HUMAN
491,24
3
862,41
1
y7
6,4
16655
CRPPVGCEELVR
5
sp|P22692|IBP4_HUMAN
491,24
3
805,39
1
y6
6,4
7047
CRPPVGCEELVR
5
sp|P22692|IBP4_HUMAN
491,24
3
645,36
1
y5
6,4
19298
CRPPVGCEELVR
5
sp|P22692|IBP4_HUMAN
491,24
3
516,31
1
y4
6,4
14093
CRPPVGCEELVR
5
sp|P22692|IBP4_HUMAN
491,24
3
387,27
1
y3
6,35
11771
CRPPVGCEELVR
5
sp|P22692|IBP4_HUMAN
491,24
3
274,19
1
y2
6,46
8168
CRPPVGCEELVR
5
sp|P22692|IBP4_HUMAN
491,24
3
578,29
2
y10
6,35
7367
CRPPVGCEELVR
5
sp|P22692|IBP4_HUMAN
491,24
3
403,20
2
y6
6,46
5605
VNGAPR
6
sp|P22692|IBP4_HUMAN
307,17
2
214,12
1
b2
1,28
227017
VNGAPR
6
sp|P22692|IBP4_HUMAN
307,17
2
220,12
2
b5
1,2
17711
VNGAPR
6
sp|P22692|IBP4_HUMAN
307,17
2
272,17
1
y2
1,16
9908
VNGAPR
6
sp|P22692|IBP4_HUMAN
307,17
2
257,64
2
y5
1,2
8484
VNGAPR
6
sp|P22692|IBP4_HUMAN
307,17
2
200,62
2
y4
1,2
3592
EDARPVPQGSCQSELHR
4
sp|P22692|IBP4_HUMAN
655,98
3
245,08
1
b2
5,45
1059
EDARPVPQGSCQSELHR
4
sp|P22692|IBP4_HUMAN
655,98
3
472,22
1
b4
5,35
1968
EDARPVPQGSCQSELHR
4
sp|P22692|IBP4_HUMAN
655,98
3
668,34
1
b6
5,45
7567
EDARPVPQGSCQSELHR
4
sp|P22692|IBP4_HUMAN
655,98
3
950,47
1
b9
5,35
908
EDARPVPQGSCQSELHR
4
sp|P22692|IBP4_HUMAN
655,98
3
519,25
2
b10
5,45
1513
EDARPVPQGSCQSELHR
4
sp|P22692|IBP4_HUMAN
655,98
3
1016,46
1
y8
5,45
757
EDARPVPQGSCQSELHR
4
sp|P22692|IBP4_HUMAN
655,98
3
929,43
1
y7
5,4
2876
EDARPVPQGSCQSELHR
4
sp|P22692|IBP4_HUMAN
655,98
3
641,34
1
y5
5,35
4389
EDARPVPQGSCQSELHR
4
sp|P22692|IBP4_HUMAN
655,98
3
312,18
1
y2
5,35
3481
EDARPVPQGSCQSELHR
4
sp|P22692|IBP4_HUMAN
655,98
3
649,80
2
y11
5,35
5449
EDARPVPQGSCQSELHR
4
sp|P22692|IBP4_HUMAN
655,98
3
537,24
2
y9
5,5
1513
EDARPVPQGSCQSELHR
4
sp|P22692|IBP4_HUMAN
655,98
3
321,17
2
y5
5,4
605
LAASQSR
7
sp|P22692|IBP4_HUMAN
366,70
2
343,20
1
b4
1,31
4692
LAASQSR
7
sp|P22692|IBP4_HUMAN
366,70
2
279,65
2
b6
1,31
45027
LAASQSR
7
sp|P22692|IBP4_HUMAN
366,70
2
262,15
1
y2
1,31
3481
LAASQSR
7
sp|P22692|IBP4_HUMAN
366,70
2
310,16
2
y6
1,26
8097
LAASQSR
7
sp|P22692|IBP4_HUMAN
366,70
2
274,64
2
y5
1,31
22173
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
928,45
2
483,18
1
b4
8,96
619
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
928,45
2
596,27
1
b5
8,96
1115
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
928,45
2
759,33
1
b6
9,08
1610
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
928,45
2
872,41
1
b7
8,96
3468
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
928,45
2
985,50
1
b8
9,04
3096
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
928,45
2
1097,58
1
y9
8,96
867
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
928,45
2
984,49
1
y8
9,04
1734
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
928,45
2
871,41
1
y7
9
4211
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
928,45
2
661,27
1
y5
8,96
2477
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
928,45
2
744,88
2
y12
9,04
743
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
619,31
3
239,11
1
b2
8,96
4211
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
619,31
3
368,16
1
b3
9
1486
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
619,31
3
483,18
1
b4
8,96
5511
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
619,31
3
596,27
1
b5
9,04
11580
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
619,31
3
759,33
1
b6
8,96
30343
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
619,31
3
872,41
1
b7
8,96
53318
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
619,31
3
985,50
1
b8
8,96
9660
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
619,31
3
380,17
2
b6
9
2353
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
619,31
3
436,71
2
b7
8,96
8174
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
619,31
3
493,25
2
b8
8,96
4830
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
619,31
3
541,78
2
b9
9,41
2477
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
619,31
3
984,49
1
y8
9
2229
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
619,31
3
871,41
1
y7
9
55981
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
619,31
3
774,36
1
y6
8,96
5202
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
619,31
3
661,27
1
y5
8,96
52141
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
619,31
3
564,22
1
y4
8,92
4582
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
619,31
3
492,75
2
y8
8,96
5264
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
619,31
3
436,21
2
y7
8,96
11147
THEDLYIIPIPNCDR
3
sp|P22692|IBP4_HUMAN
619,31
3
331,14
2
y5
8,96
10280
NGNFHPK
8
sp|P22692|IBP4_HUMAN
407,20
2
286,11
1
b3
1,41
26865
NGNFHPK
8
sp|P22692|IBP4_HUMAN
407,20
2
285,62
2
b5
1,41
21038
NGNFHPK
8
sp|P22692|IBP4_HUMAN
407,20
2
334,15
2
b6
1,36
1665
NGNFHPK
8
sp|P22692|IBP4_HUMAN
407,20
2
244,17
1
y2
1,31
1665
NGNFHPK
8
sp|P22692|IBP4_HUMAN
407,20
2
321,67
2
y5
1,36
2422
QCHPALDGQR
2
sp|P22692|IBP4_HUMAN
394,52
3
426,16
1
b3
4,96
2882
QCHPALDGQR
2
sp|P22692|IBP4_HUMAN
394,52
3
297,63
2
b5
4,96
2401
QCHPALDGQR
2
sp|P22692|IBP4_HUMAN
394,52
3
756,40
1
y7
4,96
4483
QCHPALDGQR
2
sp|P22692|IBP4_HUMAN
394,52
3
659,35
1
y6
4,91
2722
QCHPALDGQR
2
sp|P22692|IBP4_HUMAN
394,52
3
588,31
1
y5
4,96
4963
QCHPALDGQR
2
sp|P22692|IBP4_HUMAN
394,52
3
475,23
1
y4
4,96
23535
QCHPALDGQR
2
sp|P22692|IBP4_HUMAN
394,52
3
360,20
1
y3
4,96
13448
QCHPALDGQR
2
sp|P22692|IBP4_HUMAN
394,52
3
527,25
2
y9
5,02
640
QCHPALDGQR
2
sp|P22692|IBP4_HUMAN
394,52
3
378,70
2
y7
4,91
7204
CWCVDR
9
sp|P22692|IBP4_HUMAN
448,18
2
347,12
1
b2
6,46
2497
CWCVDR
9
sp|P22692|IBP4_HUMAN
448,18
2
735,32
1
y5
6,41
2573
CWCVDR
9
sp|P22692|IBP4_HUMAN
448,18
2
549,24
1
y4
6,46
14908
CWCVDR
9
sp|P22692|IBP4_HUMAN
448,18
2
389,21
1
y3
6,46
6584
CWCVDR
9
sp|P22692|IBP4_HUMAN
448,18
2
290,15
1
y2
6,46
4086
LPGGLEPK
1
sp|P22692|IBP4_HUMAN
405,74
2
211,14
1
b2
6,81
24216
LPGGLEPK
1
sp|P22692|IBP4_HUMAN
405,74
2
325,19
1
b4
6,81
16119
LPGGLEPK
1
sp|P22692|IBP4_HUMAN
405,74
2
567,31
1
b6
6,76
8778
LPGGLEPK
1
sp|P22692|IBP4_HUMAN
405,74
2
697,39
1
y7
6,76
71815
LPGGLEPK
1
sp|P22692|IBP4_HUMAN
405,74
2
600,34
1
y6
6,76
141209
LPGGLEPK
1
sp|P22692|IBP4_HUMAN
405,74
2
543,31
1
y5
6,76
17481
LPGGLEPK
1
sp|P22692|IBP4_HUMAN
405,74
2
244,17
1
y2
6,81
149987
LPGGLEPK
1
sp|P22692|IBP4_HUMAN
405,74
2
349,20
2
y7
6,76
370277
LPGGLEPK
1
sp|P22692|IBP4_HUMAN
405,74
2
243,65
2
y4
6,76
7265
GELDCHQLADSFR
10
sp|P22692|IBP4_HUMAN
516,57
3
300,16
1
b3
7,47
5764
GELDCHQLADSFR
10
sp|P22692|IBP4_HUMAN
516,57
3
575,21
1
b5
7,42
1121
GELDCHQLADSFR
10
sp|P22692|IBP4_HUMAN
516,57
3
712,27
1
b6
7,47
961
GELDCHQLADSFR
10
sp|P22692|IBP4_HUMAN
516,57
3
840,33
1
b7
7,53
6084
GELDCHQLADSFR
10
sp|P22692|IBP4_HUMAN
516,57
3
953,41
1
b8
7,42
3682
GELDCHQLADSFR
10
sp|P22692|IBP4_HUMAN
516,57
3
420,67
2
b7
7,47
3682
GELDCHQLADSFR
10
sp|P22692|IBP4_HUMAN
516,57
3
477,21
2
b8
7,42
3282
GELDCHQLADSFR
10
sp|P22692|IBP4_HUMAN
516,57
3
570,24
2
b10
7,42
961
GELDCHQLADSFR
10
sp|P22692|IBP4_HUMAN
516,57
3
973,49
1
y8
7,47
5764
GELDCHQLADSFR
10
sp|P22692|IBP4_HUMAN
516,57
3
836,43
1
y7
7,47
29618
GELDCHQLADSFR
10
sp|P22692|IBP4_HUMAN
516,57
3
708,37
1
y6
7,47
22734
GELDCHQLADSFR
10
sp|P22692|IBP4_HUMAN
516,57
3
595,28
1
y5
7,47
39705
GELDCHQLADSFR
10
sp|P22692|IBP4_HUMAN
516,57
3
524,25
1
y4
7,47
23535
GELDCHQLADSFR
10
sp|P22692|IBP4_HUMAN
516,57
3
409,22
1
y3
7,47
35862
GELDCHQLADSFR
10
sp|P22692|IBP4_HUMAN
516,57
3
322,19
1
y2
7,47
3682
GELDCHQLADSFR
10
sp|P22692|IBP4_HUMAN
516,57
3
681,32
2
y11
7,42
103024
GELDCHQLADSFR
10
sp|P22692|IBP4_HUMAN
516,57
3
624,77
2
y10
7,47
58757
GELDCHQLADSFR
10
sp|P22692|IBP4_HUMAN
516,57
3
567,26
2
y9
7,42
31860
GELDCHQLADSFR
10
sp|P22692|IBP4_HUMAN
516,57
3
487,25
2
y8
7,47
18411
GELDCHQLADSFR
10
sp|P22692|IBP4_HUMAN
516,57
3
354,69
2
y6
7,47
2401
GELDCHQLADSFR
10
sp|P22692|IBP4_HUMAN
516,57
3
298,15
2
y5
7,53
6084
GELDCHQLADSFR
10
sp|P22692|IBP4_HUMAN
516,57
3
262,63
2
y4
7,47
1601
Таблица 18: сводка площадей пиков для различных переходов для SHBG_HUMAN
Последовательность пептида
SEQ ID №
Название белка
Mz предшественника
Заряд предшественника
Mz продукта
Заряд продукта
Фрагментарный ион
Время удержания
Площадь пика rSHBG
Площадь пика объединенной сыворотки беременных
TSSSFEVR
11
sp|P04278|SHBG_HUMAN
456,72
2
811,39
1
y7
6,18
26852
96178
TSSSFEVR
11
sp|P04278|SHBG_HUMAN
456,72
2
724,36
1
y6
6,18
104140
406657
TSSSFEVR
11
sp|P04278|SHBG_HUMAN
456,72
2
637,33
1
y5
6,18
39819
152232
TSSSFEVR
11
sp|P04278|SHBG_HUMAN
456,72
2
550,30
1
y4
6,18
33489
134866
TSSSFEVR
11
sp|P04278|SHBG_HUMAN
456,72
2
403,23
1
y3
6,18
27156
91485
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
919,93
2
288,13
1
b2
9,44
28789
430926
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
919,93
2
403,16
1
b3
9,44
48399
551674
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
919,93
2
629,26
1
b5
9,44
5719
37766
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
919,93
2
686,28
1
b6
9,44
4288
48075
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
919,93
2
785,35
1
b7
9,44
19603
255484
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
919,93
2
898,43
1
b8
9,44
9799
106444
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
919,93
2
1045,50
1
b9
9,44
2959
34703
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
919,93
2
1153,59
1
y10
9,49
2043
48983
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
919,93
2
1054,52
1
y9
9,44
23075
403061
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
919,93
2
941,44
1
y8
9,44
17663
302129
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
919,93
2
794,37
1
y7
9,39
10616
212103
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
919,93
2
631,30
1
y6
9,44
13070
199732
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
919,93
2
574,28
1
y5
9,44
2040
35430
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
919,93
2
459,26
1
y4
9,49
5716
86862
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
919,93
2
358,21
1
y3
9,44
1836
39288
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
919,93
2
244,17
1
y2
9,49
11027
123399
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
919,93
2
776,37
2
y14
9,44
12561
174726
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
919,93
2
718,86
2
y13
9,44
38597
604225
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
919,93
2
287,65
2
y5
9,49
4901
88390
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
613,62
3
288,13
1
b2
9,44
8782
30219
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
613,62
3
403,16
1
b3
9,44
7759
81236
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
613,62
3
686,28
1
b6
9,44
8984
65110
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
613,62
3
785,35
1
b7
9,44
30014
161864
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
613,62
3
898,43
1
b8
9,44
12149
65219
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
613,62
3
1045,50
1
b9
9,44
20004
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
613,62
3
343,64
2
b6
9,44
22039
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
613,62
3
449,72
2
b8
9,44
13058
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
613,62
3
523,25
2
b9
9,49
10924
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
613,62
3
633,30
2
b11
9,39
27875
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
613,62
3
741,33
2
b13
9,44
23467
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
613,62
3
1054,52
1
y9
9,49
22048
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
613,62
3
941,44
1
y8
9,39
27157
111649
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
613,62
3
794,37
1
y7
9,44
43700
251500
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
613,62
3
631,30
1
y6
9,44
56356
290887
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
613,62
3
574,28
1
y5
9,39
7863
50921
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
613,62
3
459,26
1
y4
9,49
12457
66024
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
613,62
3
358,21
1
y3
9,39
8376
26955
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
613,62
3
244,17
1
y2
9,39
17667
74103
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
613,62
3
776,37
2
y14
9,49
22867
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
613,62
3
718,86
2
y13
9,44
11628
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
613,62
3
471,22
2
y8
9,44
21129
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
613,62
3
397,69
2
y7
9,39
9192
46444
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
613,62
3
316,16
2
y6
9,49
37345
TWDPEGVIFYGDTNPK
12
sp|P04278|SHBG_HUMAN
613,62
3
287,65
2
y5
9,44
17354
DDWFMLGLR
13
sp|P04278|SHBG_HUMAN
576,78
2
231,06
1
b2
10,57
2450
218844
DDWFMLGLR
13
sp|P04278|SHBG_HUMAN
576,78
2
417,14
1
b3
10,62
4085
660895
DDWFMLGLR
13
sp|P04278|SHBG_HUMAN
576,78
2
564,21
1
b4
10,67
2652
190972
DDWFMLGLR
13
sp|P04278|SHBG_HUMAN
576,78
2
695,25
1
b5
10,62
56841
DDWFMLGLR
13
sp|P04278|SHBG_HUMAN
576,78
2
922,50
1
y7
10,62
206072
DDWFMLGLR
13
sp|P04278|SHBG_HUMAN
576,78
2
736,42
1
y6
10,62
12655
1487537
DDWFMLGLR
13
sp|P04278|SHBG_HUMAN
576,78
2
589,35
1
y5
10,62
16227
1563060
DDWFMLGLR
13
sp|P04278|SHBG_HUMAN
576,78
2
458,31
1
y4
10,67
4489
681922
DDWFMLGLR
13
sp|P04278|SHBG_HUMAN
576,78
2
345,22
1
y3
10,57
7755
826140
DDWFMLGLR
13
sp|P04278|SHBG_HUMAN
576,78
2
288,20
1
y2
10,62
100631
DDWFMLGLR
13
sp|P04278|SHBG_HUMAN
576,78
2
519,27
2
y8
10,62
25104
DDWFMLGLR
13
sp|P04278|SHBG_HUMAN
576,78
2
461,75
2
y7
10,62
83186
DDWFMLGLR
13
sp|P04278|SHBG_HUMAN
384,86
3
417,14
1
b3
10,62
11229
DDWFMLGLR
13
sp|P04278|SHBG_HUMAN
384,86
3
564,21
1
b4
10,57
7349
DDWFMLGLR
13
sp|P04278|SHBG_HUMAN
384,86
3
589,35
1
y5
10,57
2450
DDWFMLGLR
13
sp|P04278|SHBG_HUMAN
384,86
3
458,31
1
y4
10,62
16538
DDWFMLGLR
13
sp|P04278|SHBG_HUMAN
384,86
3
345,22
1
y3
10,62
30524
DDWFMLGLR
13
sp|P04278|SHBG_HUMAN
384,86
3
288,20
1
y2
10,51
3880
DGRPEIQLHNHWAQLTVGAGPR
14
sp|P04278|SHBG_HUMAN
818,09
3
329,16
1
b3
7,84
5943
24455
DGRPEIQLHNHWAQLTVGAGPR
14
sp|P04278|SHBG_HUMAN
818,09
3
555,25
1
b5
7,72
8188
DGRPEIQLHNHWAQLTVGAGPR
14
sp|P04278|SHBG_HUMAN
818,09
3
796,39
1
b7
7,84
3326
13408
DGRPEIQLHNHWAQLTVGAGPR
14
sp|P04278|SHBG_HUMAN
818,09
3
909,48
1
b8
7,84
5227
15675
DGRPEIQLHNHWAQLTVGAGPR
14
sp|P04278|SHBG_HUMAN
818,09
3
1046,54
1
b9
7,72
12351
DGRPEIQLHNHWAQLTVGAGPR
14
sp|P04278|SHBG_HUMAN
818,09
3
1160,58
1
b10
7,84
5462
11163
DGRPEIQLHNHWAQLTVGAGPR
14
sp|P04278|SHBG_HUMAN
818,09
3
523,77
2
b9
7,72
8555
DGRPEIQLHNHWAQLTVGAGPR
14
sp|P04278|SHBG_HUMAN
818,09
3
580,79
2
b10
7,84
4630
24587
DGRPEIQLHNHWAQLTVGAGPR
14
sp|P04278|SHBG_HUMAN
818,09
3
649,32
2
b11
7,78
8555
24107
DGRPEIQLHNHWAQLTVGAGPR
14
sp|P04278|SHBG_HUMAN
818,09
3
742,36
2
b12
7,9
4753
20193
DGRPEIQLHNHWAQLTVGAGPR
14
sp|P04278|SHBG_HUMAN
818,09
3
777,88
2
b13
7,9
8312
19238
DGRPEIQLHNHWAQLTVGAGPR
14
sp|P04278|SHBG_HUMAN
818,09
3
841,91
2
b14
7,72
8551
DGRPEIQLHNHWAQLTVGAGPR
14
sp|P04278|SHBG_HUMAN
818,09
3
898,45
2
b15
7,84
6656
29217
DGRPEIQLHNHWAQLTVGAGPR
14
sp|P04278|SHBG_HUMAN
818,09
3
1062,54
2
b19
7,84
3328
6649
DGRPEIQLHNHWAQLTVGAGPR
14
sp|P04278|SHBG_HUMAN
818,09
3
1155,63
1
y11
7,9
11995
32069
DGRPEIQLHNHWAQLTVGAGPR
14
sp|P04278|SHBG_HUMAN
818,09
3
969,55
1
y10
7,84
16386
47509
DGRPEIQLHNHWAQLTVGAGPR
14
sp|P04278|SHBG_HUMAN
818,09
3
898,51
1
y9
7,84
9025
26601
DGRPEIQLHNHWAQLTVGAGPR
14
sp|P04278|SHBG_HUMAN
818,09
3
770,45
1
y8
7,78
6410
16278
DGRPEIQLHNHWAQLTVGAGPR
14
sp|P04278|SHBG_HUMAN
818,09
3
657,37
1
y7
7,84
4990
21850
DGRPEIQLHNHWAQLTVGAGPR
14
sp|P04278|SHBG_HUMAN
818,09
3
556,32
1
y6
7,9
3089
8193
DGRPEIQLHNHWAQLTVGAGPR
14
sp|P04278|SHBG_HUMAN
818,09
3
457,25
1
y5
7,9
3324
9848
DGRPEIQLHNHWAQLTVGAGPR
14
sp|P04278|SHBG_HUMAN
818,09
3
329,19
1
y3
7,9
7600
11648
DGRPEIQLHNHWAQLTVGAGPR
14
sp|P04278|SHBG_HUMAN
818,09
3
1062,56
2
y19
7,78
3802
9572
DGRPEIQLHNHWAQLTVGAGPR
14
sp|P04278|SHBG_HUMAN
818,09
3
646,85
2
y12
7,9
3328
7497
DGRPEIQLHNHWAQLTVGAGPR
14
sp|P04278|SHBG_HUMAN
818,09
3
329,19
2
y7
7,84
10685
5422
WHQVEVK
15
sp|P04278|SHBG_HUMAN
463,25
2
324,15
1
b2
5,82
95843
143834
WHQVEVK
15
sp|P04278|SHBG_HUMAN
463,25
2
452,20
1
b3
5,88
24228
30155
WHQVEVK
15
sp|P04278|SHBG_HUMAN
463,25
2
551,27
1
b4
5,82
10215
16269
WHQVEVK
15
sp|P04278|SHBG_HUMAN
463,25
2
680,32
1
b5
5,88
31354
35986
WHQVEVK
15
sp|P04278|SHBG_HUMAN
463,25
2
779,38
1
b6
5,82
8316
12828
WHQVEVK
15
sp|P04278|SHBG_HUMAN
463,25
2
739,41
1
y6
5,82
29929
37874
WHQVEVK
15
sp|P04278|SHBG_HUMAN
463,25
2
602,35
1
y5
5,82
57721
77194
WHQVEVK
15
sp|P04278|SHBG_HUMAN
463,25
2
246,18
1
y2
5,82
91450
141340
WHQVEVK
15
sp|P04278|SHBG_HUMAN
463,25
2
370,21
2
y6
5,82
88601
90134
WHQVEVK
15
sp|P04278|SHBG_HUMAN
309,17
3
324,15
1
b2
5,82
89310
94424
WHQVEVK
15
sp|P04278|SHBG_HUMAN
309,17
3
452,20
1
b3
5,88
82658
107490
WHQVEVK
15
sp|P04278|SHBG_HUMAN
309,17
3
551,27
1
b4
5,82
57008
47029
WHQVEVK
15
sp|P04278|SHBG_HUMAN
309,17
3
340,66
2
b5
5,82
47270
48456
WHQVEVK
15
sp|P04278|SHBG_HUMAN
309,17
3
602,35
1
y5
5,82
20904
24944
WHQVEVK
15
sp|P04278|SHBG_HUMAN
309,17
3
474,29
1
y4
5,82
43114
35632
WHQVEVK
15
sp|P04278|SHBG_HUMAN
309,17
3
246,18
1
y2
5,82
284438
306895
WHQVEVK
15
sp|P04278|SHBG_HUMAN
309,17
3
370,21
2
y6
5,82
431957
434922
WHQVEVK
15
sp|P04278|SHBG_HUMAN
309,17
3
301,68
2
y5
5,82
32539
42866
WHQVEVK
15
sp|P04278|SHBG_HUMAN
309,17
3
237,65
2
y4
5,82
24000
22570
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
945,46
2
261,09
1
b2
9,56
9976
176237
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
945,46
2
318,11
1
b3
9,5
4160
68533
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
945,46
2
433,14
1
b4
9,56
11047
235634
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
945,46
2
520,17
1
b5
9,44
89909
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
945,46
2
619,24
1
b6
9,56
21139
230412
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
945,46
2
732,32
1
b7
9,56
13304
236332
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
945,46
2
845,41
1
b8
9,56
12592
166750
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
945,46
2
974,45
1
b9
9,56
5708
94539
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
945,46
2
1073,52
1
b10
9,44
58323
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
945,46
2
1188,55
1
b11
9,44
32055
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
945,46
2
260,59
2
b5
9,44
29113
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
945,46
2
1158,60
1
y10
9,5
20897
448830
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
945,46
2
1045,52
1
y9
9,56
32183
563430
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
945,46
2
916,47
1
y8
9,56
29571
345839
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
945,46
2
817,41
1
y7
9,62
26010
438470
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
945,46
2
702,38
1
y6
9,56
13669
211762
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
945,46
2
645,36
1
y5
9,5
4037
32785
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
945,46
2
516,31
1
y4
9,56
5585
68764
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
945,46
2
387,27
1
y3
9,56
5459
81359
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
945,46
2
288,20
1
y2
9,5
9506
114023
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
630,64
3
261,09
1
b2
9,56
6062
51794
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
630,64
3
433,14
1
b4
9,5
9495
42761
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
630,64
3
619,24
1
b6
9,38
12348
65809
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
630,64
3
732,32
1
b7
9,5
21619
85170
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
630,64
3
974,45
1
b9
9,5
22572
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
630,64
3
366,67
2
b7
9,56
4985
27676
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
630,64
3
423,21
2
b8
9,44
36344
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
630,64
3
487,73
2
b9
9,5
20069
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
630,64
3
1045,52
1
y9
9,56
9148
74821
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
630,64
3
916,47
1
y8
9,56
21496
135854
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
630,64
3
817,41
1
y7
9,5
57831
380769
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
630,64
3
702,38
1
y6
9,56
46795
236942
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
630,64
3
645,36
1
y5
9,56
8320
42640
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
630,64
3
516,31
1
y4
9,62
21264
102732
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
630,64
3
387,27
1
y3
9,5
9267
84073
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
630,64
3
288,20
1
y2
9,32
12234
53912
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
630,64
3
685,88
2
y12
9,44
17945
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
630,64
3
458,74
2
y8
9,44
19110
MEGDSVLLEVDGEEVLR
16
sp|P04278|SHBG_HUMAN
630,64
3
323,18
2
y5
9,56
24717
QVSGPLTSK
17
sp|P04278|SHBG_HUMAN
458,76
2
228,13
1
b2
5,86
11214
57191
QVSGPLTSK
17
sp|P04278|SHBG_HUMAN
458,76
2
788,45
1
y8
5,86
2990
19062
QVSGPLTSK
17
sp|P04278|SHBG_HUMAN
458,76
2
689,38
1
y7
5,86
61053
282713
QVSGPLTSK
17
sp|P04278|SHBG_HUMAN
458,76
2
602,35
1
y6
5,86
22679
104538
QVSGPLTSK
17
sp|P04278|SHBG_HUMAN
458,76
2
545,33
1
y5
5,86
27536
123604
QVSGPLTSK
17
sp|P04278|SHBG_HUMAN
458,76
2
448,28
1
y4
5,86
5233
26420
QVSGPLTSK
17
sp|P04278|SHBG_HUMAN
458,76
2
335,19
1
y3
5,86
29403
111637
QVSGPLTSK
17
sp|P04278|SHBG_HUMAN
458,76
2
234,14
1
y2
5,86
21802
79995
QVSGPLTSK
17
sp|P04278|SHBG_HUMAN
458,76
2
345,20
2
y7
5,86
14455
58815
QVSGPLTSK
17
sp|P04278|SHBG_HUMAN
458,76
2
301,68
2
y6
5,86
2990
18189
QVSGPLTSK
17
sp|P04278|SHBG_HUMAN
458,76
2
273,17
2
y5
5,86
16948
89586
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
721,43
2
298,21
1
b3
10,65
6981
63802
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
721,43
2
355,23
1
b4
10,72
4236
56059
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
721,43
2
412,26
1
b5
10,65
25923
402833
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
721,43
2
525,34
1
b6
10,65
41881
404680
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
721,43
2
638,42
1
b7
10,59
14960
144040
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
721,43
2
785,49
1
b8
10,65
10219
105535
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
721,43
2
1144,65
1
y11
10,65
22931
282840
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
721,43
2
1087,63
1
y10
10,65
10465
140548
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
721,43
2
1030,60
1
y9
10,72
6483
125959
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
721,43
2
917,52
1
y8
10,65
49362
594214
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
721,43
2
804,44
1
y7
10,65
76280
973633
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
721,43
2
657,37
1
y6
10,65
93730
1204642
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
721,43
2
560,32
1
y5
10,59
3242
53952
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
721,43
2
489,28
1
y4
10,65
49330
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
721,43
2
402,25
1
y3
10,65
23177
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
721,43
2
288,20
1
y2
10,65
18686
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
721,43
2
629,37
2
y12
10,65
5481
73517
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
721,43
2
572,83
2
y11
10,72
11466
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
721,43
2
402,72
2
y7
10,65
20181
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
721,43
2
329,19
2
y6
10,65
21538
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
721,43
2
245,14
2
y4
10,65
6485
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
481,29
3
298,21
1
b3
10,65
15331
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
481,29
3
412,26
1
b5
10,65
13956
193878
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
481,29
3
525,34
1
b6
10,65
24679
323212
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
481,29
3
638,42
1
b7
10,65
11589
110026
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
481,29
3
785,49
1
b8
10,59
2494
16200
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
481,29
3
319,72
2
b7
10,65
14451
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
481,29
3
393,25
2
b8
10,59
3745
12199
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
481,29
3
917,52
1
y8
10,65
21931
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
481,29
3
804,44
1
y7
10,65
7478
96064
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
481,29
3
657,37
1
y6
10,65
79020
937227
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
481,29
3
560,32
1
y5
10,65
19940
364330
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
481,29
3
489,28
1
y4
10,65
14459
185782
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
481,29
3
402,25
1
y3
10,59
4236
37877
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
481,29
3
288,20
1
y2
10,59
15698
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
481,29
3
402,72
2
y7
10,59
9968
81983
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
481,29
3
329,19
2
y6
10,65
67299
722053
IALGGLLFPASNLR
18
sp|P04278|SHBG_HUMAN
481,29
3
245,14
2
y4
10,72
16079
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
662,38
2
211,14
1
b2
9,72
142292
756192
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
662,38
2
324,23
1
b3
9,72
160489
866724
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
662,38
2
423,30
1
b4
9,66
194131
842302
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
662,38
2
212,15
2
b4
9,66
3491
131435
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
662,38
2
296,20
2
b6
9,66
4983
68644
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
662,38
2
1113,61
1
y10
9,72
24667
97571
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
662,38
2
1000,52
1
y9
9,66
27161
108785
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
662,38
2
901,46
1
y8
9,72
469991
1998965
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
662,38
2
804,40
1
y7
9,66
9969
74638
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
662,38
2
733,37
1
y6
9,66
9723
52218
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
662,38
2
620,28
1
y5
9,66
9966
75133
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
662,38
2
505,26
1
y4
9,72
28658
150379
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
662,38
2
605,83
2
y11
9,66
39377
199487
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
662,38
2
500,77
2
y9
9,66
11339
51218
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
662,38
2
451,23
2
y8
9,66
47221
214689
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
662,38
2
310,64
2
y5
9,66
48970
270761
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
441,92
3
211,14
1
b2
9,72
3988
4863
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
441,92
3
324,23
1
b3
9,72
3235
20432
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
441,92
3
423,30
1
b4
9,66
12709
38129
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
441,92
3
591,39
1
b6
9,72
20185
50468
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
441,92
3
704,47
1
b7
9,72
4615
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
441,92
3
260,68
2
b5
9,66
2987
5981
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
441,92
3
733,37
1
y6
9,66
3988
9096
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
441,92
3
620,28
1
y5
9,66
23677
67284
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
441,92
3
505,26
1
y4
9,66
19692
45854
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
441,92
3
448,23
1
y3
9,66
2243
8470
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
441,92
3
367,19
2
y6
9,72
5484
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
441,92
3
310,64
2
y5
9,59
4736
6356
LPLVPALDGCLR
19
sp|P04278|SHBG_HUMAN
441,92
3
253,13
2
y4
9,72
3491
8842
DSWLDK
20
sp|P04278|SHBG_HUMAN
382,18
2
203,07
1
b2
7,1
36632
DSWLDK
20
sp|P04278|SHBG_HUMAN
382,18
2
389,15
1
b3
7,1
2990
DSWLDK
20
sp|P04278|SHBG_HUMAN
382,18
2
648,34
1
y5
7,1
10216
DSWLDK
20
sp|P04278|SHBG_HUMAN
382,18
2
561,30
1
y4
7,1
59060
DSWLDK
20
sp|P04278|SHBG_HUMAN
382,18
2
375,22
1
y3
7,1
75758
DSWLDK
20
sp|P04278|SHBG_HUMAN
382,18
2
262,14
1
y2
7,1
71394
DSWLDK
20
sp|P04278|SHBG_HUMAN
382,18
2
324,67
2
y5
7,1
11711
DSWLDK
20
sp|P04278|SHBG_HUMAN
382,18
2
281,16
2
y4
7,1
7726
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
665,85
2
200,10
1
b2
6,84
126856
265599
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
665,85
2
329,15
1
b3
6,84
225724
470069
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
665,85
2
442,23
1
b4
6,8
64901
134361
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
665,85
2
529,26
1
b5
6,84
20563
41566
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
665,85
2
600,30
1
b6
6,88
15019
27276
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
665,85
2
687,33
1
b7
6,84
7335
19406
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
665,85
2
758,37
1
b8
6,84
8226
19758
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
665,85
2
1131,60
1
y11
6,84
80998
171464
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
665,85
2
1002,56
1
y10
6,84
83503
195346
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
665,85
2
889,47
1
y9
6,84
365275
865394
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
665,85
2
802,44
1
y8
6,84
170577
346969
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
665,85
2
731,40
1
y7
6,84
184606
420174
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
665,85
2
644,37
1
y6
6,84
99233
217518
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
665,85
2
573,34
1
y5
6,84
204455
471407
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
665,85
2
476,28
1
y4
6,8
9298
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
665,85
2
375,24
1
y3
6,8
9116
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
665,85
2
288,20
1
y2
6,84
7689
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
665,85
2
566,30
2
y11
6,8
7063
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
665,85
2
501,78
2
y10
6,84
8043
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
444,24
3
200,10
1
b2
6,84
4738
70979
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
444,24
3
329,15
1
b3
6,8
8223
26541
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
444,24
3
442,23
1
b4
6,84
9478
15644
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
444,24
3
529,26
1
b5
6,8
6614
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
444,24
3
600,30
1
b6
6,8
5900
23599
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
444,24
3
687,33
1
b7
6,75
2859
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
444,24
3
344,17
2
b7
6,84
2682
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
444,24
3
731,40
1
y7
6,84
7869
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
444,24
3
644,37
1
y6
6,88
12335
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
444,24
3
573,34
1
y5
6,84
138213
82159
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
444,24
3
476,28
1
y4
6,8
33434
19037
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
444,24
3
375,24
1
y3
6,88
18686
11795
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
444,24
3
288,20
1
y2
6,8
139993
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
444,24
3
445,24
2
y9
6,84
1789
8675
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
444,24
3
401,72
2
y8
6,75
75276
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
444,24
3
322,69
2
y6
6,84
4828
QAEISASAPTSLR
21
sp|P04278|SHBG_HUMAN
444,24
3
287,17
2
y5
6,8
99236
33965
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
248,07
1
b2
9,75
42019
101416
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
363,10
1
b3
9,7
85912
184040
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
462,17
1
b4
9,7
38534
77277
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
591,21
1
b5
9,7
40944
79511
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
678,24
1
b6
9,7
23602
59566
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
792,28
1
b7
9,75
48097
86937
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
946,36
1
b9
9,75
58289
107231
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
1059,44
1
b10
9,7
53017
96052
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
445,17
2
b8
9,75
12607
47034
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
473,68
2
b9
9,7
50063
86740
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
530,22
2
b10
9,7
36838
58848
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
603,76
2
b11
9,7
11442
36577
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
660,30
2
b12
9,75
15913
24059
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
757,35
2
b14
9,7
25030
42298
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
1132,57
1
y10
9,7
110857
213116
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
1035,52
1
y9
9,7
34689
70918
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
978,50
1
y8
9,66
13408
29510
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
877,45
1
y7
9,75
21816
44261
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
749,39
1
y6
9,7
44877
76365
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
678,36
1
y5
9,7
30398
67608
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
549,31
1
y4
9,75
29679
77350
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
288,20
1
y2
9,7
24322
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
878,97
2
y16
9,75
10731
14845
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
671,86
2
y12
9,75
8226
15831
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
615,32
2
y11
9,7
754906
1053051
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
566,79
2
y10
9,7
83146
139155
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
375,20
2
y6
9,7
10192
19233
SCDVESNPGIFLPPGTQAEFNLR
22
sp|P04278|SHBG_HUMAN
850,08
3
275,16
2
y4
9,79
19761
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
229,12
1
b2
10,33
5965
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
326,17
1
b3
10,46
11667
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
454,23
1
b4
10,4
8742
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
551,28
1
b5
10,4
6984
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
688,34
1
b6
10,33
16485
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
759,38
1
b7
10,33
19657
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
888,42
1
b8
10,4
38546
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
1171,55
1
b10
10,4
8234
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
276,14
2
b5
10,52
7732
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
586,28
2
b10
10,46
6088
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
695,33
2
b12
10,46
5961
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
738,85
2
b13
10,4
8373
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
852,90
2
b15
10,4
6596
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
909,45
2
b16
10,27
7093
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
937,96
2
b17
10,4
12049
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
994,50
2
b18
10,4
6720
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
963,55
1
y9
10,46
8243
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
892,51
1
y8
10,4
28277
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
745,45
1
y7
10,4
17877
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
658,41
1
y6
10,33
6455
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
545,33
1
y5
10,33
15214
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
430,30
1
y4
10,4
51478
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
317,22
1
y3
10,4
24348
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
260,20
1
y2
10,4
11287
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
1010,04
2
y18
10,33
67074
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
953,50
2
y17
10,4
226206
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
904,97
2
y16
10,33
7357
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
840,94
2
y15
10,4
137962
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
792,41
2
y14
10,4
11410
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
688,37
2
y12
10,33
12934
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
575,32
2
y10
10,33
3557
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
373,23
2
y7
10,33
16856
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
1067,55
2
215,65
2
y4
10,27
6851
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
712,04
3
229,12
1
b2
10,33
54641
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
712,04
3
888,42
1
b8
10,33
15214
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
712,04
3
1149,63
1
y10
10,33
11157
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
712,04
3
963,55
1
y9
10,33
3928
87243
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
712,04
3
892,51
1
y8
10,38
8027
165351
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
712,04
3
745,45
1
y7
10,33
6318
159006
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
712,04
3
658,41
1
y6
10,33
67207
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
712,04
3
545,33
1
y5
10,33
2393
59596
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
712,04
3
430,30
1
y4
10,38
3584
86992
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
712,04
3
317,22
1
y3
10,33
2390
60223
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
712,04
3
260,20
1
y2
10,33
19028
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
712,04
3
953,50
2
y17
10,38
6828
77604
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
712,04
3
840,94
2
y15
10,38
9905
125911
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
712,04
3
792,41
2
y14
10,33
17238
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
712,04
3
688,37
2
y12
10,4
8880
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
712,04
3
623,85
2
y11
10,38
4778
57062
DIPQPHAEPWAFSLDLGLK
23
sp|P04278|SHBG_HUMAN
712,04
3
446,76
2
y8
10,33
28150
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
312,23
1
b3
12,14
14018
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
399,26
1
b4
12,09
13357
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
486,29
1
b5
12,09
10441
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
543,31
1
b6
12,14
15143
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
630,35
1
b7
12,17
3930
14951
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
687,37
1
b8
12,13
3925
26904
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
784,42
1
b9
12,09
7900
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
841,44
1
b10
12,13
3930
10719
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
954,53
1
b11
12,13
2903
25772
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
1069,55
1
b12
12,13
4955
16747
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
1182,64
1
b13
12,04
4099
31707
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
535,28
2
b12
12,04
5641
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
936,55
2
b20
12,04
13640
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
1190,79
1
y11
12,13
17593
91335
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
1093,73
1
y10
12,09
8089
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
980,65
1
y9
12,09
21070
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
881,58
1
y8
12,08
18961
80043
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
768,50
1
y7
12,13
18444
102065
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
711,48
1
y6
12,08
3074
21355
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
598,39
1
y5
12,08
47057
292259
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
501,34
1
y4
12,09
3760
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
388,26
1
y3
12,08
2732
21073
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
260,20
1
y2
12,04
3074
11477
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
1087,64
2
y22
12,08
3077
21073
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
1031,10
2
y21
12,09
15240
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
987,59
2
y20
12,09
8371
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
944,07
2
y19
12,13
3415
11761
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
915,56
2
y18
12,14
8558
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
872,04
2
y17
12,13
4609
19566
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
843,53
2
y16
12,04
15899
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
766,49
2
y14
12,14
4141
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
652,44
2
y12
12,14
8088
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
1186,71
2
441,29
2
y8
12,09
6774
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
312,23
1
b3
12,13
42888
65949
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
399,26
1
b4
12,13
21029
43367
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
486,29
1
b5
12,07
7286
7151
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
543,31
1
b6
12,18
9370
12223
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
630,35
1
b7
12,13
18326
24266
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
687,37
1
b8
12,13
19156
21728
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
784,42
1
b9
12,13
11660
23424
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
841,44
1
b10
12,13
16347
20317
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
954,53
1
b11
12,13
15821
19379
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
1069,55
1
b12
12,07
46738
71574
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
1182,64
1
b13
12,13
47885
64624
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
421,22
2
b10
12,13
5101
13732
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
477,77
2
b11
12,18
5206
6681
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
535,28
2
b12
12,13
11661
18625
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
591,82
2
b13
12,07
30187
42141
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
640,35
2
b14
12,13
52254
63769
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
696,89
2
b15
12,13
16655
17121
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
746,42
2
b16
12,13
8535
10155
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
936,55
2
b20
12,07
5000
8561
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
1190,79
1
y11
12,13
51215
73554
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
1093,73
1
y10
12,07
23529
31977
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
980,65
1
y9
12,07
51626
77042
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
881,58
1
y8
12,13
153018
225474
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
768,50
1
y7
12,07
369345
512663
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
711,48
1
y6
12,13
64333
87008
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
598,39
1
y5
12,13
329370
537778
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
501,34
1
y4
12,13
10510
16372
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
388,26
1
y3
12,13
30707
60384
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
260,20
1
y2
12,07
18529
33308
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
872,04
2
y17
12,13
4166
6771
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
766,49
2
y14
12,07
12077
14396
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
652,44
2
y12
12,07
7494
10257
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
595,90
2
y11
12,07
33522
42701
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
547,37
2
y10
12,13
3954
4984
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
441,29
2
y8
12,13
7701
15425
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
356,24
2
y6
12,07
6871
8751
VVLSSGSGPGLDLPLVLGLPLQLK
24
sp|P04278|SHBG_HUMAN
791,48
3
299,70
2
y5
12,07
7701
9689
VVLSQGSK
25
sp|P04278|SHBG_HUMAN
409,24
2
312,23
1
b3
5,25
14004
16657
VVLSQGSK
25
sp|P04278|SHBG_HUMAN
409,24
2
584,34
1
b6
5,22
9128
VVLSQGSK
25
sp|P04278|SHBG_HUMAN
409,24
2
718,41
1
y7
5,25
101369
154048
VVLSQGSK
25
sp|P04278|SHBG_HUMAN
409,24
2
619,34
1
y6
5,25
860577
1157529
VVLSQGSK
25
sp|P04278|SHBG_HUMAN
409,24
2
506,26
1
y5
5,21
168229
273074
VVLSQGSK
25
sp|P04278|SHBG_HUMAN
409,24
2
419,22
1
y4
5,25
40477
58906
VVLSQGSK
25
sp|P04278|SHBG_HUMAN
409,24
2
291,17
1
y3
5,25
56277
98421
VVLSQGSK
25
sp|P04278|SHBG_HUMAN
409,24
2
234,14
1
y2
5,3
18448
21652
LDVDQALNR
26
sp|P04278|SHBG_HUMAN
522,28
2
229,12
1
b2
7,22
175928
LDVDQALNR
26
sp|P04278|SHBG_HUMAN
522,28
2
328,19
1
b3
7,22
30537
50598
LDVDQALNR
26
sp|P04278|SHBG_HUMAN
522,28
2
443,21
1
b4
7,22
24914
LDVDQALNR
26
sp|P04278|SHBG_HUMAN
522,28
2
571,27
1
b5
7,18
8188
LDVDQALNR
26
sp|P04278|SHBG_HUMAN
522,28
2
642,31
1
b6
7,06
27320
LDVDQALNR
26
sp|P04278|SHBG_HUMAN
522,28
2
869,44
1
b8
7,06
22459
LDVDQALNR
26
sp|P04278|SHBG_HUMAN
522,28
2
435,22
2
b8
7,06
24039
LDVDQALNR
26
sp|P04278|SHBG_HUMAN
522,28
2
930,46
1
y8
7,22
66345
99621
LDVDQALNR
26
sp|P04278|SHBG_HUMAN
522,28
2
815,44
1
y7
7,22
128600
224769
LDVDQALNR
26
sp|P04278|SHBG_HUMAN
522,28
2
716,37
1
y6
7,22
176273
329489
LDVDQALNR
26
sp|P04278|SHBG_HUMAN
522,28
2
601,34
1
y5
7,22
112569
239451
LDVDQALNR
26
sp|P04278|SHBG_HUMAN
522,28
2
473,28
1
y4
7,18
84667
185259
LDVDQALNR
26
sp|P04278|SHBG_HUMAN
522,28
2
402,25
1
y3
7,18
69746
100254
LDVDQALNR
26
sp|P04278|SHBG_HUMAN
522,28
2
289,16
1
y2
7,22
55177
96879
LDVDQALNR
26
sp|P04278|SHBG_HUMAN
522,28
2
408,22
2
y7
7,18
21442
LDVDQALNR
26
sp|P04278|SHBG_HUMAN
522,28
2
358,69
2
y6
7,18
4997
LDVDQALNR
26
sp|P04278|SHBG_HUMAN
522,28
2
201,63
2
y3
7,22
10549
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
1155,20
2
256,17
1
b3
12,18
79381
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
1155,20
2
369,25
1
b4
12,12
20134
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
1155,20
2
466,30
1
b5
12,23
11181
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
1155,20
2
676,44
1
b7
12,12
14124
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
1155,20
2
733,46
1
b8
12,07
12965
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
1155,20
2
846,54
1
b9
12,18
19293
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
1155,20
2
917,58
1
b10
12,18
14550
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
1155,20
2
282,18
2
b6
12,07
7805
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
1155,20
2
1069,59
1
y9
12,12
6538
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
1155,20
2
842,46
1
y7
12,07
8531
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
1155,20
2
656,38
1
y6
12,23
14766
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
1155,20
2
585,35
1
y5
12,18
9813
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
1155,20
2
457,25
1
y4
12,18
45762
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
1155,20
2
1063,14
2
y20
12,12
8012
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
1155,20
2
1027,62
2
y19
12,07
17082
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
1155,20
2
971,08
2
y18
12,12
214978
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
1155,20
2
922,55
2
y17
12,12
7694
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
1155,20
2
478,28
2
y8
12,12
7702
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
256,17
1
b3
12,07
842606
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
369,25
1
b4
12,12
82661
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
466,30
1
b5
12,07
17819
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
563,36
1
b6
12,02
10122
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
733,46
1
b8
12,12
9487
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
282,18
2
b6
12,07
48813
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
367,23
2
b8
12,07
11705
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
1182,67
1
y10
12,07
25519
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
1069,59
1
y9
12,12
53562
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
955,55
1
y8
12,12
29098
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
842,46
1
y7
12,07
164685
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
656,38
1
y6
12,12
180607
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
585,35
1
y5
12,07
93202
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
457,25
1
y4
12,12
285196
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
360,20
1
y3
12,12
8859
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
232,14
1
y2
12,12
13702
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
1027,62
2
y19
12,07
7692
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
971,08
2
y18
12,07
616149
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
922,55
2
y17
12,12
82239
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
874,02
2
y16
12,12
25832
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
817,48
2
y15
12,07
64945
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
788,97
2
y14
12,12
12867
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
732,43
2
y13
12,07
48079
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
696,91
2
y12
12,07
72642
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
648,38
2
y11
12,12
238495
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
591,84
2
y10
12,12
11281
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
535,30
2
y9
12,12
19196
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
478,28
2
y8
12,07
92887
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
421,74
2
y7
12,12
29947
ALALPPLGLAPLLNLWAKPQGR
27
sp|P04278|SHBG_HUMAN
770,47
3
293,18
2
y5
12,02
12438
SHEIWTHSCPQSPGNGTDASH
28
sp|P04278|SHBG_HUMAN
768,99
3
467,22
1
b4
7,56
7050
SHEIWTHSCPQSPGNGTDASH
28
sp|P04278|SHBG_HUMAN
768,99
3
653,30
1
b5
7,46
43014
SHEIWTHSCPQSPGNGTDASH
28
sp|P04278|SHBG_HUMAN
768,99
3
446,21
2
b7
7,61
9066
SHEIWTHSCPQSPGNGTDASH
28
sp|P04278|SHBG_HUMAN
768,99
3
682,30
2
b11
7,41
6441
SHEIWTHSCPQSPGNGTDASH
28
sp|P04278|SHBG_HUMAN
768,99
3
774,34
2
b13
7,46
6544
SHEIWTHSCPQSPGNGTDASH
28
sp|P04278|SHBG_HUMAN
768,99
3
942,39
1
y10
7,46
4031
SHEIWTHSCPQSPGNGTDASH
28
sp|P04278|SHBG_HUMAN
768,99
3
701,28
1
y7
7,41
8864
SHEIWTHSCPQSPGNGTDASH
28
sp|P04278|SHBG_HUMAN
768,99
3
530,22
1
y5
7,41
6044
SHEIWTHSCPQSPGNGTDASH
28
sp|P04278|SHBG_HUMAN
768,99
3
429,17
1
y4
7,46
13502
SHEIWTHSCPQSPGNGTDASH
28
sp|P04278|SHBG_HUMAN
768,99
3
314,15
1
y3
7,41
19243
SHEIWTHSCPQSPGNGTDASH
28
sp|P04278|SHBG_HUMAN
768,99
3
243,11
1
y2
7,51
46537
SHEIWTHSCPQSPGNGTDASH
28
sp|P04278|SHBG_HUMAN
768,99
3
664,27
2
y13
7,41
9468
SHEIWTHSCPQSPGNGTDASH
28
sp|P04278|SHBG_HUMAN
768,99
3
428,18
2
y9
7,51
13303
SHEIWTHSCPQSPGNGTDASH
28
sp|P04278|SHBG_HUMAN
768,99
3
351,15
2
y7
7,46
6446
SHEIWTHSCPQSPGNGTDASH
28
sp|P04278|SHBG_HUMAN
768,99
3
215,09
2
y4
7,51
11478
Различные изоформы
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
706,89
2
211,14
1
b2
10,93
86522
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
706,89
2
282,18
1
b3
10,88
73637
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
706,89
2
411,22
1
b4
10,98
8856
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
706,89
2
682,38
1
b7
10,93
19344
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
706,89
2
840,45
1
b9
10,88
21252
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
706,89
2
937,50
1
b10
10,88
28413
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
706,89
2
341,69
2
b7
10,88
17422
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
706,89
2
385,21
2
b8
10,93
9772
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
706,89
2
573,34
1
y5
10,88
6857
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
706,89
2
476,28
1
y4
10,93
197840
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
706,89
2
288,20
1
y2
10,93
6351
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
706,89
2
445,24
2
y9
10,88
23775
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
706,89
2
287,17
2
y5
10,83
12993
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
471,60
3
211,14
1
b2
10,93
148281
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
471,60
3
282,18
1
b3
10,93
23973
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
471,60
3
411,22
1
b4
10,93
17728
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
471,60
3
524,31
1
b5
10,93
3629
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
471,60
3
682,38
1
b7
10,93
8865
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
471,60
3
840,45
1
b9
10,93
1614
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
471,60
3
937,50
1
b10
10,93
1914
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
471,60
3
341,69
2
b7
10,88
8765
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
471,60
3
385,21
2
b8
10,88
4734
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
471,60
3
469,25
2
b10
10,88
4633
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
471,60
3
563,29
2
b12
10,88
3025
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
471,60
3
644,37
1
y6
10,93
5434
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
471,60
3
476,28
1
y4
10,88
333632
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
471,60
3
288,20
1
y2
10,88
5642
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
471,60
3
650,35
2
y13
10,88
12993
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
471,60
3
566,30
2
y11
10,93
1813
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
471,60
3
445,24
2
y9
10,88
36867
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
471,60
3
366,21
2
y7
10,93
4734
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
471,60
3
322,69
2
y6
10,93
3424
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
471,60
3
287,17
2
y5
10,88
19746
LPAEISASAPTSLR
29
sp|P04278-5|SHBG_HUMAN
471,60
3
238,64
2
y4
10,83
9876
TLPPLFA
30
sp|P04278-2|SHBG_HUMAN
379,73
2
312,19
1
b3
5,99
99852
TLPPLFA
30
sp|P04278-2|SHBG_HUMAN
379,73
2
350,21
1
y3
5,99
830722
TLPPLFA
30
sp|P04278-2|SHBG_HUMAN
379,73
2
237,12
1
y2
5,99
1013802
TLPPLFA
30
sp|P04278-2|SHBG_HUMAN
379,73
2
272,66
2
y5
5,99
2760482
GEDSSTSFCLNGLWAQGQR
31
sp|P04278-4|SHBG_HUMAN
704,98
3
389,13
1
b4
10,84
355452
GEDSSTSFCLNGLWAQGQR
31
sp|P04278-4|SHBG_HUMAN
704,98
3
476,16
1
b5
10,84
71118
GEDSSTSFCLNGLWAQGQR
31
sp|P04278-4|SHBG_HUMAN
704,98
3
577,21
1
b6
10,84
15469
GEDSSTSFCLNGLWAQGQR
31
sp|P04278-4|SHBG_HUMAN
704,98
3
289,11
2
b6
10,84
29667
GEDSSTSFCLNGLWAQGQR
31
sp|P04278-4|SHBG_HUMAN
704,98
3
332,62
2
b7
10,84
471567
GEDSSTSFCLNGLWAQGQR
31
sp|P04278-4|SHBG_HUMAN
704,98
3
406,16
2
b8
10,84
26243
GEDSSTSFCLNGLWAQGQR
31
sp|P04278-4|SHBG_HUMAN
704,98
3
486,17
2
b9
10,84
28271
GEDSSTSFCLNGLWAQGQR
31
sp|P04278-4|SHBG_HUMAN
704,98
3
542,72
2
b10
10,84
11031
GEDSSTSFCLNGLWAQGQR
31
sp|P04278-4|SHBG_HUMAN
704,98
3
599,74
2
b11
10,84
15214
GEDSSTSFCLNGLWAQGQR
31
sp|P04278-4|SHBG_HUMAN
704,98
3
628,25
2
b12
10,84
27636
GEDSSTSFCLNGLWAQGQR
31
sp|P04278-4|SHBG_HUMAN
704,98
3
813,35
2
b15
10,84
12932
GEDSSTSFCLNGLWAQGQR
31
sp|P04278-4|SHBG_HUMAN
704,98
3
858,46
1
y7
10,84
12672
GEDSSTSFCLNGLWAQGQR
31
sp|P04278-4|SHBG_HUMAN
704,98
3
745,37
1
y6
10,84
32451
GEDSSTSFCLNGLWAQGQR
31
sp|P04278-4|SHBG_HUMAN
704,98
3
488,26
1
y4
10,84
16233
GEDSSTSFCLNGLWAQGQR
31
sp|P04278-4|SHBG_HUMAN
704,98
3
360,20
1
y3
10,84
54774
GEDSSTSFCLNGLWAQGQR
31
sp|P04278-4|SHBG_HUMAN
704,98
3
819,40
2
y14
10,84
40433
GEDSSTSFCLNGLWAQGQR
31
sp|P04278-4|SHBG_HUMAN
704,98
3
651,82
2
y11
10,9
31186
GEDSSTSFCLNGLWAQGQR
31
sp|P04278-4|SHBG_HUMAN
704,98
3
515,26
2
y9
10,78
15092
GEDSSTSFCLNGLWAQGQR
31
sp|P04278-4|SHBG_HUMAN
704,98
3
458,24
2
y8
10,78
16351
GEDSSTSFCLNGLWAQGQR
31
sp|P04278-4|SHBG_HUMAN
704,98
3
373,19
2
y6
10,84
39431
Таблица 19: белки с измененными уровнями в сыворотке в течение 17-25 недель GA в PTB образцах
Белок
Изменение
Функциональная категория
THRB
повыш.
коагуляция/ответ острой фазы
VTNC
повыш.
клеточная адгезия/ответ острой фазы
HEMO
повыш.
транспорт гема/ответ острой фазы
FETUA
повыш.
воспаление/ответ острой фазы
LBP
повыш.
врожденный иммунитет/ответ острой фазы
IBP4
повыш.
регуляция факторов роста
CD14
повыш.
врожденный иммунитет
HABP2
повыш.
клеточная адгезия/миграция
INHBC
повыш.
регуляция факторов роста
CFAB
повыш.
комплемент/ответ острой фазы
ICAM1
повыш.
клеточная адгезия/миграция
IC1
повыш.
комплемент/ответ острой фазы
APOH
повыш.
коагуляция/аутоиммунность
B2MG
повыш.
MHC/иммунитет
C1S
повыш.*
комплемент
APOE
повыш.*
метаболизм холестерина
APOC3
повыш.*
метаболизм триглицеридов
PEDF
повыш.*
Ангиогенез
CATD
повыш.*
ECM ремоделирование/клеточная миграция
INHBE
повыш.*
регуляция факторов роста
IBP6
повыш.*
регуляция факторов роста
PRG2
пониж.
регуляция факторов роста
SHBG
пониж.
воспаление/метаболизм стероидов
GELS
пониж.
связывание актина/ответ острой фазы
PSG4
пониж.*
регуляция факторов роста
*Дополнительные белки, ограниченные неделями 19-21 GA при PTB.
Таблица 20: 44 белка, прошедшие аналитические фильтры, которые находились под повышающей или понижающей регуляцией при sPTD в сравнении с полносрочными контролями
Uniprot ID
Короткое название
Название белка
A2GL_HUMAN
LRG1
Богатый лейцином α2-гликопротеин
AFAM_HUMAN
AFM
Афамин
ANGT_HUMAN
AGT
Ангиотензиноген
APOC3_HUMAN
APOC3
Аполипопротеин C-III
APOH_HUMAN
APOH
β2-гликопротеин 1
B2MG_HUMAN
B2M
β2-микроглобулин
BGH3_HUMAN
TGFBI
Индуцированный трансформирующим фактором роста β белок ig-h3
CATD_HUMAN
CTSD
Катепсин D
CBPN_HUMAN
CPN1
Каталитическая цепь карбоксипептидазы N
CD14_HUMAN
CD14
Антиген дифференцировки моноцитов CD14
CFAB_HUMAN
CFB
Фактор B комплемента
CHL1_HUMAN
CHL1
Белок, подобный молекуле адгезии нервных клеток L1
CO5_HUMAN
C5
Комплемент C5
CO6_HUMAN
C6
Компонент комплемента C6
CO8A_HUMAN
C8A
Компонент комплемента C8, цепь α
CRIS3_HUMAN
CRISP3
Богатый цистеином секреторный белок 3
ENPP2_HUMAN
ENPP2
Член семейства эктонуклеотидпирофосфатаз/фосфодиэстераз 2
F13B_HUMAN
F13B
Фактор свертывания XIII, цепь B
FBLN3_HUMAN
EFEMP1
EGF-содержащий фибулин-подобный белок внеклеточного матрикса 1
FETUA_HUMAN
AHSG
α2-HS-гликопротеин
HABP2_HUMAN
HABP2
Связывающий гиалуроновую кислоту белок 2
HEMO_HUMAN
HPX
Гемопексин
HLAG_HUMAN
HLA-G*
Антиген гистосовместимости HLA I класса, α цепь G
IBP2_HUMAN
IGFBP2
Связывающий инсулиноподобный фактор роста белок 2
IBP3_HUMAN
IGFBP3
Связывающий инсулиноподобный фактор роста белок 3
IBP4_HUMAN
IGFBP4
Связывающий инсулиноподобный фактор роста белок 4
INHBC_HUMAN
INHBC
Ингибин β, цепь C
ITIH3_HUMAN
ITIH3
Тяжелая цепь интер-альфа-ингибитора трипсина H3
ITIH4_HUMAN
ITIH4
Тяжелая цепь интер-альфа-ингибитора трипсина H4, N-конец
ITIH4_HUMAN
ITIH4
Тяжелая цепь интер-альфа-ингибитора трипсина H4, C-конец
KNG1_HUMAN
KNG1
Кининоген-1
LBP_HUMAN
LBP
Липополисахаридсвязывающий белок
LYAM3_HUMAN
SELP
P-селектин
PAPP1_HUMAN
PAPPA
Паппализин-1
PEDF_HUMAN
SERPINF1
Фактор пигментного эпителия
PGRP2_HUMAN
PGLYRP2
N-ацетилмурамоил-L-аланинамидаза
PRG2_HUMAN
PRG2
Протеогликан костного мозга
PSG11_HUMAN
PSG11
Специфичный β1-гликопротеин беременности 11
PSG2_HUMAN
PSG2
Специфичный β1-гликопротеин беременности 2
PSG9_HUMAN
PSG9
Специфичный β1-гликопротеин беременности 9
SHBG_HUMAN
SHBG
Связывающий половые гормоны глобулин
TENX_HUMAN
TNXB
Тенасцин-X
TIE1_HUMAN
TIE1
Рецептор тирозинпротеинкиназы Tie-1
VTNC_HUMAN
VTN
Витронектин
* пептидный суррогат для HLA-G не был уникальным для этого белка.
Таблица 21
Белок
Переход
SEQ ID №
Тип перехода
Истощенный MARS14 белок
SIS переход
SEQ ID №
A1AG1_HUMAN
NWGLSVYADKPETTK_570.3_301.1
32
количественн.
Истощен
IS_NWGLSVYADKPETTK_573.0_301.1
32
A1AG1_HUMAN
NWGLSVYADKPETTK_570.3_818.4
32
качественн.
Истощен
IS_NWGLSVYADKPETTK_573.0_826.4
32
A1AT_HUMAN
LSITGTYDLK_555.8_696.4
33
качественн.
Истощен
IS_LSITGTYDLK_559.8_704.4
33
A1AT_HUMAN
LSITGTYDLK_555.8_797.4
33
количественн.
Истощен
IS_LSITGTYDLK_559.8_805.4
33
A2GL_HUMAN
DLLLPQPDLR_590.3_229.1
34
качественн.
IS_DLLLPQPDLR_595.3_229.1
34
A2GL_HUMAN
DLLLPQPDLR_590.3_725.4
34
количественн.
IS_DLLLPQPDLR_595.3_735.4
34
A2GL_HUMAN
LQVLGK_329.2_204.1
35
качественн.
A2GL_HUMAN
LQVLGK_329.2_416.3
35
количественн.
A2MG_HUMAN
LHTEAQIQEEGTVVELTGR_704.0_674.4
36
качественн.
Истощен
IS_LHTEAQIQEEGTVVELTGR_707.4_680.3
36
A2MG_HUMAN
LHTEAQIQEEGTVVELTGR_704.0_680.3
36
количественн.
Истощен
IS_LHTEAQIQEEGTVVELTGR_707.4_684.4
36
AFAM_HUMAN
DADPDTFFAK_563.8_302.1
37
качественн.
IS_DADPDTFFAK_567.8_302.1
37
AFAM_HUMAN
DADPDTFFAK_563.8_825.4
37
количественн.
IS_DADPDTFFAK_567.8_833.4
37
AFAM_HUMAN
HFQNLGK_422.2_285.1
38
количественн.
IS_HFQNLGK_426.2_285.1
38
AFAM_HUMAN
HFQNLGK_422.2_527.2
38
качественн.
IS_HFQNLGK_426.2_527.2
38
ALBU_HUMAN
LVTDLTK_395.2_213.2
39
качественн.
Истощен
IS_LVTDLTK_399.2_213.2
39
ALBU_HUMAN
LVTDLTK_395.2_577.3
39
количественн.
Истощен
IS_LVTDLTK_399.2_585.3
39
ALS_HUMAN
IRPHTFTGLSGLR_485.6_432.3
40
количественн.
IS_IRPHTFTGLSGLR_488.9_442.3
40
ALS_HUMAN
IRPHTFTGLSGLR_485.6_545.3
40
качественн.
IS_IRPHTFTGLSGLR_488.9_555.3
40
ALS_HUMAN
LEYLLLSR_503.8_447.3
41
качественн.
ALS_HUMAN
LEYLLLSR_503.8_745.4
41
количественн.
ANGT_HUMAN
DPTFIPAPIQAK_433.2_461.2
42
качественн.
IS_DPTFIPAPIQAK_435.9_461.2
42
ANGT_HUMAN
DPTFIPAPIQAK_433.2_556.3
42
количественн.
IS_DPTFIPAPIQAK_435.9_564.4
42
ANGT_HUMAN
SLDFTELDVAAEK_719.4_316.2
43
количественн.
ANGT_HUMAN
SLDFTELDVAAEK_719.4_874.5
43
качественн.
APOA1_HUMAN
AKPALEDLR_506.8_288.2
44
качественн.
Истощен
IS_AKPALEDLR_511.8_298.2
44
APOA1_HUMAN
AKPALEDLR_506.8_813.5
44
количественн.
Истощен
IS_AKPALEDLR_511.8_823.5
44
APOA2_HUMAN
SPELQAEAK_486.8_659.4
45
качественн.
Истощен
IS_SPELQAEAK_490.8_667.4
45
APOA2_HUMAN
SPELQAEAK_486.8_788.4
45
количественн.
Истощен
IS_SPELQAEAK_490.8_796.4
45
APOC3_HUMAN
DYWSTVK_449.7_347.2
46
качественн.
APOC3_HUMAN
DYWSTVK_449.7_620.3
46
количественн.
APOC3_HUMAN
GWVTDGFSSLK_598.8_854.4
47
количественн.
IS_GWVTDGFSSLK_602.8_862.4
47
APOC3_HUMAN
GWVTDGFSSLK_598.8_953.5
47
качественн.
IS_GWVTDGFSSLK_602.8_961.5
47
APOH_HUMAN
ATVVYQGER_511.8_652.3
48
количественн.
IS_ATVVYQGER_516.8_662.3
48
APOH_HUMAN
ATVVYQGER_511.8_751.4
48
качественн.
IS_ATVVYQGER_516.8_761.4
48
APOH_HUMAN
EHSSLAFWK_552.8_267.1
49
качественн.
APOH_HUMAN
EHSSLAFWK_552.8_838.4
49
количественн.
B2MG_HUMAN
VEHSDLSFSK_383.5_234.1
50
качественн.
IS_VEHSDLSFSK_386.2_242.2
50
B2MG_HUMAN
VEHSDLSFSK_383.5_468.2
50
количественн.
IS_VEHSDLSFSK_386.2_476.3
50
B2MG_HUMAN
VNHVTLSQPK_374.9_244.2
51
качественн.
IS_VNHVTLSQPK_377.6_252.2
51
B2MG_HUMAN
VNHVTLSQPK_374.9_459.3
51
количественн.
IS_VNHVTLSQPK_377.6_467.3
51
BGH3_HUMAN
LTLLAPLNSVFK_658.4_804.5
52
количественн.
IS_LTLLAPLNSVFK_662.4_812.5
52
BGH3_HUMAN
LTLLAPLNSVFK_658.4_875.5
52
качественн.
IS_LTLLAPLNSVFK_662.4_883.5
52
BGH3_HUMAN
VLTDELK_409.2_605.3
53
количественн.
BGH3_HUMAN
VLTDELK_409.2_718.4
53
качественн.
C163A_HUMAN
INPASLDK_429.2_462.3
54
качественн.
IS_INPASLDK_433.2_470.3
54
C163A_HUMAN
INPASLDK_429.2_630.4
54
количественн.
IS_INPASLDK_433.2_638.4
54
C1QB_HUMAN
VPGLYYFTYHASSR_554.3_420.2
55
количественн.
IS_VPGLYYFTYHASSR_557.6_430.2
55
C1QB_HUMAN
VPGLYYFTYHASSR_554.3_720.3
55
качественн.
IS_VPGLYYFTYHASSR_557.6_730.4
55
CAH1_HUMAN
GGPFSDSYR_493.2_627.3
56
количественн.
IS_GGPFSDSYR_498.2_637.3
56
CAH1_HUMAN
GGPFSDSYR_493.2_774.3
56
качественн.
IS_GGPFSDSYR_498.2_784.3
56
CATD_HUMAN
VGFAEAAR_410.7_517.3
57
качественн.
IS_VGFAEAAR_415.7_527.3
57
CATD_HUMAN
VGFAEAAR_410.7_721.4
57
количественн.
IS_VGFAEAAR_415.7_731.4
57
CATD_HUMAN
VSTLPAITLK_521.8_642.4
58
количественн.
IS_VSTLPAITLK_525.8_650.4
58
CATD_HUMAN
VSTLPAITLK_521.8_856.6
58
качественн.
IS_VSTLPAITLK_525.8_864.6
58
CBPN_HUMAN
EALIQFLEQVHQGIK_585.0_526.3
59
качественн.
IS_EALIQFLEQVHQGIK_587.7_530.3
59
CBPN_HUMAN
EALIQFLEQVHQGIK_585.0_720.4
59
количественн.
IS_EALIQFLEQVHQGIK_587.7_724.4
59
CBPN_HUMAN
NNANGVDLNR_543.8_229.1
60
количественн.
IS_NNANGVDLNR_548.8_229.1
60
CBPN_HUMAN
NNANGVDLNR_543.8_858.4
60
качественн.
IS_NNANGVDLNR_548.8_868.5
60
CD14_HUMAN
LTVGAAQVPAQLLVGALR_889.0_416.3
61
количественн.
IS_LTVGAAQVPAQLLVGALR_894.0_426.3
61
CD14_HUMAN
LTVGAAQVPAQLLVGALR_889.0_628.4
61
качественн.
IS_LTVGAAQVPAQLLVGALR_894.0_638.4
61
CD14_HUMAN
SWLAELQQWLKPGLK_599.7_274.1
62
количественн.
IS_SWLAELQQWLKPGLK_602.3_274.1
62
CD14_HUMAN
SWLAELQQWLKPGLK_599.7_670.4
62
качественн.
IS_SWLAELQQWLKPGLK_602.3_674.4
62
CFAB_HUMAN
VSEADSSNADWVTK_754.9_347.2
63
количественн.
CFAB_HUMAN
VSEADSSNADWVTK_754.9_533.3
63
качественн.
CFAB_HUMAN
YGLVTYATYPK_638.3_334.2
64
качественн.
IS_YGLVTYATYPK_642.3_334.2
64
CFAB_HUMAN
YGLVTYATYPK_638.3_843.4
64
количественн.
IS_YGLVTYATYPK_642.3_851.4
64
CHL1_HUMAN
TAVTANLDIR_537.3_288.2
65
качественн.
CHL1_HUMAN
TAVTANLDIR_537.3_802.4
65
количественн.
CHL1_HUMAN
VIAVNEVGR_478.8_574.3
66
качественн.
IS_VIAVNEVGR_483.8_584.3
66
CHL1_HUMAN
VIAVNEVGR_478.8_744.4
66
количественн.
IS_VIAVNEVGR_483.8_754.4
66
CLUS_HUMAN
ASSIIDELFQDR_697.4_678.4
67
качественн.
IS_ASSIIDELFQDR_702.4_688.4
67
CLUS_HUMAN
ASSIIDELFQDR_697.4_922.4
67
количественн.
IS_ASSIIDELFQDR_702.4_932.4
67
CLUS_HUMAN
LFDSDPITVTVPVEVSR_937.5_1086.6
68
количественн.
IS_LFDSDPITVTVPVEVSR_942.5_1096.6
68
CLUS_HUMAN
LFDSDPITVTVPVEVSR_937.5_985.6
68
качественн.
IS_LFDSDPITVTVPVEVSR_942.5_995.6
68
CO3_HUMAN
IHWESASLLR_606.3_251.2
69
количественн.
Истощен
IS_IHWESASLLR_611.3_251.2
69
CO3_HUMAN
IHWESASLLR_606.3_437.2
69
качественн.
Истощен
IS_IHWESASLLR_611.3_437.2
69
CO5_HUMAN
TLLPVSKPEIR_418.3_288.2
70
качественн.
IS_TLLPVSKPEIR_421.6_298.2
70
CO5_HUMAN
TLLPVSKPEIR_418.3_514.3
70
количественн.
IS_TLLPVSKPEIR_421.6_524.3
70
CO5_HUMAN
VFQFLEK_455.8_276.2
71
качественн.
IS_VFQFLEK_459.8_284.2
71
CO5_HUMAN
VFQFLEK_455.8_811.4
71
количественн.
IS_VFQFLEK_459.8_819.4
71
CO6_HUMAN
ALNHLPLEYNSALYSR_621.0_538.3
72
количественн.
IS_ALNHLPLEYNSALYSR_624.3_548.3
72
CO6_HUMAN
ALNHLPLEYNSALYSR_621.0_696.4
72
качественн.
IS_ALNHLPLEYNSALYSR_624.3_706.4
72
CO6_HUMAN
SEYGAALAWEK_612.8_788.4
73
качественн.
CO6_HUMAN
SEYGAALAWEK_612.8_845.5
73
количественн.
CO8A_HUMAN
SLLQPNK_400.2_358.2
74
качественн.
IS_SLLQPNK_404.2_366.2
74
CO8A_HUMAN
SLLQPNK_400.2_599.4
74
количественн.
IS_SLLQPNK_404.2_607.4
74
CO8A_HUMAN
YHFEALADTGISSEFYDNANDLLSK_940.8_761.4
75
количественн.
CO8A_HUMAN
YHFEALADTGISSEFYDNANDLLSK_940.8_874.5
75
качественн.
CO8B_HUMAN
QALEEFQK_496.8_551.3
76
качественн.
IS_QALEEFQK_500.8_559.3
76
CO8B_HUMAN
QALEEFQK_496.8_680.3
76
количественн.
IS_QALEEFQK_500.8_688.3
76
CO8B_HUMAN
SGFSFGFK_438.7_585.3
77
количественн.
CO8B_HUMAN
SGFSFGFK_438.7_732.4
77
качественн.
CRIS3_HUMAN
AVSPPAR_349.2_258.1
78
качественн.
IS_AVSPPAR_354.2_258.1
78
CRIS3_HUMAN
AVSPPAR_349.2_343.2
78
количественн.
IS_AVSPPAR_354.2_353.2
78
CRIS3_HUMAN
YEDLYSNCK_596.3_784.4
79
качественн.
IS_YEDLYSNCK_600.3_792.4
79
CRIS3_HUMAN
YEDLYSNCK_596.3_899.4
79
количественн.
IS_YEDLYSNCK_600.3_907.4
79
CSH_HUMAN*
AHQLAIDTYQEFEETYIPK_766.0_521.3
80
качественн.
IS_AHQLAIDTYQEFEETYIPK_768.7_521.3
80
CSH_HUMAN*
AHQLAIDTYQEFEETYIPK_766.0_634.4
80
количественн.
IS_AHQLAIDTYQEFEETYIPK_768.7_634.4
80
CSH_HUMAN*
ISLLLIESWLEPVR_834.5_371.2
81
качественн.
IS_ISLLLIESWLEPVR_839.5_381.2
81
CSH_HUMAN*
ISLLLIESWLEPVR_834.5_500.3
81
количественн.
IS_ISLLLIESWLEPVR_839.5_510.3
81
ENPP2_HUMAN
TEFLSNYLTNVDDITLVPGTLGR_846.8_600.3
82
количественн.
IS_TEFLSNYLTNVDDITLVPGTLGR_850.1_610.4
82
ENPP2_HUMAN
TEFLSNYLTNVDDITLVPGTLGR_846.8_699.4
82
качественн.
IS_TEFLSNYLTNVDDITLVPGTLGR_850.1_709.4
82
ENPP2_HUMAN
TYLHTYESEI_628.3_1124.5
83
количественн.
IS_TYLHTYESEI_631.8_1124.5
83
ENPP2_HUMAN
TYLHTYESEI_628.3_908.4
83
качественн.
IS_TYLHTYESEI_631.8_908.4
83
F13B_HUMAN
GDTYPAELYITGSILR_885.0_1332.8
84
количественн.
IS_GDTYPAELYITGSILR_890.0_1342.8
84
F13B_HUMAN
GDTYPAELYITGSILR_885.0_274.1
84
качественн.
IS_GDTYPAELYITGSILR_890.0_274.1
84
F13B_HUMAN
IAQYYYTFK_598.8_395.2
85
качественн.
F13B_HUMAN
IAQYYYTFK_598.8_884.4
85
количественн.
FBLN1_HUMAN
TGYYFDGISR_589.8_694.4
86
качественн.
IS_TGYYFDGISR_594.8_704.4
86
FBLN1_HUMAN
TGYYFDGISR_589.8_857.4
86
количественн.
IS_TGYYFDGISR_594.8_867.4
86
FBLN3_HUMAN
IPSNPSHR_303.2_496.3
87
количественн.
IS_IPSNPSHR_306.5_506.3
87
FBLN3_HUMAN
IPSNPSHR_303.2_610.3
87
качественн.
IS_IPSNPSHR_306.5_620.3
87
FETUA_HUMAN
FSVVYAK_407.2_381.2
88
качественн.
IS_FSVVYAK_411.2_389.2
88
FETUA_HUMAN
FSVVYAK_407.2_579.4
88
количественн.
IS_FSVVYAK_411.2_587.4
88
FETUA_HUMAN
HTLNQIDEVK_598.8_951.5
89
качественн.
IS_HTLNQIDEVK_602.8_951.5
89
FETUA_HUMAN
HTLNQIDEVK_598.8_958.5
89
количественн.
IS_HTLNQIDEVK_602.8_966.5
89
FIBA_HUMAN
ESSSHHPGIAEFPSR_546.6_353.7
90
качественн.
Истощен
IS_ESSSHHPGIAEFPSR_549.9_358.7
90
FIBA_HUMAN
ESSSHHPGIAEFPSR_546.6_502.2
90
количественн.
Истощен
IS_ESSSHHPGIAEFPSR_549.9_502.2
90
FIBB_HUMAN
QGFGNVATNTDGK_654.8_319.2
91
качественн.
Истощен
IS_QGFGNVATNTDGK_658.8_327.2
91
FIBB_HUMAN
QGFGNVATNTDGK_654.8_706.3
91
количественн.
Истощен
IS_QGFGNVATNTDGK_658.8_714.4
91
HABP2_HUMAN
FLNWIK_410.7_560.3
92
количественн.
IS_FLNWIK_414.7_568.3
92
HABP2_HUMAN
FLNWIK_410.7_673.4
92
качественн.
IS_FLNWIK_414.7_681.4
92
HEMO_HUMAN
NFPSPVDAAFR_610.8_775.4
93
качественн.
IS_NFPSPVDAAFR_615.8_785.4
93
HEMO_HUMAN
NFPSPVDAAFR_610.8_959.5
93
количественн.
IS_NFPSPVDAAFR_615.8_969.5
93
HEMO_HUMAN
SGAQATWTELPWPHEK_613.3_510.3
94
качественн.
HEMO_HUMAN
SGAQATWTELPWPHEK_613.3_793.4
94
количественн.
HLAG_HUMAN*
WAAVVVPSGEEQR_714.4_428.2
95
качественн.
IS_WAAVVVPSGEEQR_719.4_428.2
95
HLAG_HUMAN*
WAAVVVPSGEEQR_714.4_802.4
95
количественн.
IS_WAAVVVPSGEEQR_719.4_812.4
95
HPT_HUMAN
TEGDGVYTLNNEK_720.3_403.2
96
качественн.
Истощен
IS_TEGDGVYTLNNEK_724.3_403.2
96
HPT_HUMAN
TEGDGVYTLNNEK_720.3_881.4
96
количественн.
Истощен
IS_TEGDGVYTLNNEK_724.3_889.5
96
IBP1_HUMAN
VVESLAK_373.2_547.3
97
количественн.
IS_VVESLAK_377.2_555.3
97
IBP1_HUMAN
VVESLAK_373.2_646.4
97
качественн.
IS_VVESLAK_377.2_654.4
97
IBP2_HUMAN
LIQGAPTIR_484.8_227.2
98
качественн.
IS_LIQGAPTIR_489.8_227.2
98
IBP2_HUMAN
LIQGAPTIR_484.8_742.4
98
количественн.
IS_LIQGAPTIR_489.8_752.4
98
IBP3_HUMAN
FLNVLSPR_473.3_472.3
99
качественн.
IS_FLNVLSPR_478.3_482.3
99
IBP3_HUMAN
FLNVLSPR_473.3_685.4
99
количественн.
IS_FLNVLSPR_478.3_695.4
99
IBP3_HUMAN
YGQPLPGYTTK_612.8_666.3
100
качественн.
IS_YGQPLPGYTTK_616.8_674.4
100
IBP3_HUMAN
YGQPLPGYTTK_612.8_876.5
100
количественн.
IS_YGQPLPGYTTK_616.8_884.5
100
IBP4_HUMAN
QCHPALDGQR_394.5_360.2
2
качественн.
IS_QCHPALDGQR_397.9_370.2
2
IBP4_HUMAN
QCHPALDGQR_394.5_475.2
2
количественн.
IS_QCHPALDGQR_397.9_485.2
2
IBP6_HUMAN
GAQTLYVPNCDHR_510.9_312.2
101
качественн.
IS_GAQTLYVPNCDHR_514.2_322.2
101
IBP6_HUMAN
GAQTLYVPNCDHR_510.9_637.8
101
количественн.
IS_GAQTLYVPNCDHR_514.2_642.8
101
IBP6_HUMAN
HLDSVLQQLQTEVYR_610.3_667.3
102
качественн.
IS_HLDSVLQQLQTEVYR_613.7_677.3
102
IBP6_HUMAN
HLDSVLQQLQTEVYR_610.3_795.4
102
количественн.
IS_HLDSVLQQLQTEVYR_613.7_805.4
102
IGF2_HUMAN
GIVEECCFR_585.3_771.3
103
качественн.
IS_GIVEECCFR_590.3_781.3
103
IGF2_HUMAN
GIVEECCFR_585.3_900.3
103
количественн.
IS_GIVEECCFR_590.3_910.3
103
IGF2_HUMAN
SCDLALLETYCATPAK_906.9_1040.5
104
качественн.
IGF2_HUMAN
SCDLALLETYCATPAK_906.9_1153.6
104
количественн.
IGHG3_HUMAN
ALPAPIEK_419.8_327.7
105
количественн.
Истощен
IS_ALPAPIEK_423.8_331.7
105
IGHG3_HUMAN
ALPAPIEK_419.8_654.4
105
качественн.
Истощен
IS_ALPAPIEK_423.8_662.4
105
IGHM_HUMAN
GFPSVLR_388.2_286.2
106
количественн.
Истощен
IS_GFPSVLR_393.2_291.2
106
IGHM_HUMAN
GFPSVLR_388.2_571.4
106
качественн.
Истощен
IS_GFPSVLR_393.2_581.4
106
INHBC_HUMAN
LDFHFSSDR_375.2_448.2
107
качественн.
IS_LDFHFSSDR_378.5_453.2
107
INHBC_HUMAN
LDFHFSSDR_375.2_611.3
107
количественн.
IS_LDFHFSSDR_378.5_621.3
107
IS_Recon
IS_ASSILAT_662.4_313.1
108
качественн.
IS_Recon
IS_ASSILAT_662.4_359.2
108
количественн.
IS_Recon
IS_ELWFSDDPDVTK_726.3_559.3
109
количественн.
IS_Recon
IS_ELWFSDDPDVTK_726.3_876.4
109
качественн.
IS_Recon
IS_NVDQSLLELHK_432.6_397.3
110
количественн.
IS_Recon
IS_NVDQSLLELHK_432.6_639.4
110
качественн.
ITIH3_HUMAN
ALDLSLK_380.2_185.1
111
качественн.
IS_ALDLSLK_384.2_185.1
111
ITIH3_HUMAN
ALDLSLK_380.2_575.3
111
количественн.
IS_ALDLSLK_384.2_583.4
111
ITIH4_HUMAN
ILDDLSPR_464.8_587.3
112
качественн.
IS_ILDDLSPR_469.8_597.3
112
ITIH4_HUMAN
ILDDLSPR_464.8_702.3
112
количественн.
IS_ILDDLSPR_469.8_712.4
112
ITIH4_HUMAN
NPLVWVHASPEHVVVTR_647.4_325.2
113
количественн.
IS_NPLVWVHASPEHVVVTR_650.7_325.2
113
ITIH4_HUMAN
NPLVWVHASPEHVVVTR_647.4_936.5
113
качественн.
IS_NPLVWVHASPEHVVVTR_650.7_946.5
113
ITIH4_HUMAN
QLGLPGPPDVPDHAAYHPF_676.7_263.1
114
количественн.
IS_QLGLPGPPDVPDHAAYHPF_680.0_273.2
114
ITIH4_HUMAN
QLGLPGPPDVPDHAAYHPF_676.7_299.2
114
качественн.
IS_QLGLPGPPDVPDHAAYHPF_680.0_299.2
114
ITIH4_HUMAN
VRPQQLVK_484.3_609.4
115
количественн.
ITIH4_HUMAN
VRPQQLVK_484.3_722.4
115
качественн.
KNG1_HUMAN
DIPTNSPELEETLTHTITK_713.7_756.4
116
количественн.
IS_DIPTNSPELEETLTHTITK_716.4_760.4
116
KNG1_HUMAN
DIPTNSPELEETLTHTITK_713.7_799.9
116
качественн.
IS_DIPTNSPELEETLTHTITK_716.4_803.9
116
KNG1_HUMAN
QVVAGLNFR_502.3_606.3
117
качественн.
IS_QVVAGLNFR_507.3_616.3
117
KNG1_HUMAN
QVVAGLNFR_502.3_677.4
117
количественн.
IS_QVVAGLNFR_507.3_687.4
117
LBP_HUMAN
ITGFLKPGK_320.9_301.2
118
количественн.
IS_ITGFLKPGK_323.5_309.2
118
LBP_HUMAN
ITGFLKPGK_320.9_429.3
118
качественн.
IS_ITGFLKPGK_323.5_437.3
118
LBP_HUMAN
ITLPDFTGDLR_624.3_288.2
119
качественн.
IS_ITLPDFTGDLR_629.3_298.2
119
LBP_HUMAN
ITLPDFTGDLR_624.3_920.5
119
количественн.
IS_ITLPDFTGDLR_629.3_930.5
119
LYAM3_HUMAN*
SYYWIGIR_529.3_644.4
120
качественн.
IS_SYYWIGIR_534.3_654.4
120
LYAM3_HUMAN*
SYYWIGIR_529.3_807.5
120
количественн.
IS_SYYWIGIR_534.3_817.5
120
NCAM1_HUMAN
GLGEISAASEFK_604.8_357.2
121
качественн.
IS_GLGEISAASEFK_608.8_357.2
121
NCAM1_HUMAN
GLGEISAASEFK_604.8_739.4
121
количественн.
IS_GLGEISAASEFK_608.8_747.4
121
PAPP1_HUMAN
DIPHWLNPTR_416.9_373.2
122
количественн.
IS_DIPHWLNPTR_420.2_383.2
122
PAPP1_HUMAN
DIPHWLNPTR_416.9_600.4
122
качественн.
IS_DIPHWLNPTR_420.2_610.4
122
PAPP1_HUMAN
LDGSTHLNIFFAK_488.3_739.4
123
количественн.
PAPP1_HUMAN
LDGSTHLNIFFAK_488.3_852.5
123
качественн.
PEDF_HUMAN
LQSLFDSPDFSK_692.3_329.2
124
качественн.
IS_LQSLFDSPDFSK_696.4_329.2
124
PEDF_HUMAN
LQSLFDSPDFSK_692.3_942.4
124
количественн.
IS_LQSLFDSPDFSK_696.4_950.4
124
PEDF_HUMAN
TVQAVLTVPK_528.3_428.3
125
качественн.
IS_TVQAVLTVPK_532.3_432.3
125
PEDF_HUMAN
TVQAVLTVPK_528.3_855.5
125
количественн.
IS_TVQAVLTVPK_532.3_863.5
125
PGRP2_HUMAN
AGLLRPDYALLGHR_518.0_369.2
126
количественн.
IS_AGLLRPDYALLGHR_521.3_379.2
126
PGRP2_HUMAN
AGLLRPDYALLGHR_518.0_595.4
126
качественн.
IS_AGLLRPDYALLGHR_521.3_605.4
126
PGRP2_HUMAN
DGSPDVTTADIGANTPDATK_973.5_531.3
127
количественн.
PGRP2_HUMAN
DGSPDVTTADIGANTPDATK_973.5_844.4
127
качественн.
PRDX2_HUMAN
GLFIIDGK_431.8_319.2
128
качественн.
IS_GLFIIDGK_435.8_327.2
128
PRDX2_HUMAN
GLFIIDGK_431.8_545.3
128
количественн.
IS_GLFIIDGK_435.8_553.3
128
PRG2_HUMAN
WNFAYWAAHQPWSR_607.3_545.3
129
количественн.
IS_WNFAYWAAHQPWSR_610.6_555.3
129
PRG2_HUMAN
WNFAYWAAHQPWSR_607.3_673.3
129
качественн.
IS_WNFAYWAAHQPWSR_610.6_683.3
129
PSG1_HUMAN
DLYHYITSYVVDGEIIIYGPAYSGR_955.5_650.3
130
качественн.
PSG1_HUMAN
DLYHYITSYVVDGEIIIYGPAYSGR_955.5_707.3
130
количественн.
PSG1_HUMAN
FQLPGQK_409.2_276.1
131
количественн.
IS_FQLPGQK_413.2_276.1
131
PSG1_HUMAN
FQLPGQK_409.2_429.2
131
качественн.
IS_FQLPGQK_413.2_437.3
131
PSG11_HUMAN
LFIPQITPK_528.8_261.2
132
качественн.
IS_LFIPQITPK_532.8_261.2
132
PSG11_HUMAN
LFIPQITPK_528.8_683.4
132
количественн.
IS_LFIPQITPK_532.8_691.4
132
PSG2_HUMAN
IHPSYTNYR_384.2_338.2
133
качественн.
IS_IHPSYTNYR_387.5_348.2
133
PSG2_HUMAN
IHPSYTNYR_384.2_452.2
133
количественн.
IS_IHPSYTNYR_387.5_462.2
133
PSG3_HUMAN
VSAPSGTGHLPGLNPL_758.9_229.2
134
количественн.
IS_VSAPSGTGHLPGLNPL_762.4_236.2
134
PSG3_HUMAN
VSAPSGTGHLPGLNPL_758.9_610.4
134
качественн.
IS_VSAPSGTGHLPGLNPL_762.4_617.4
134
PSG9_HUMAN
DVLLLVHNLPQNLPGYFWYK_810.4_328.2
135
качественн.
IS_DVLLLVHNLPQNLPGYFWYK_813.1_328.2
135
PSG9_HUMAN
DVLLLVHNLPQNLPGYFWYK_810.4_960.5
135
количественн.
IS_DVLLLVHNLPQNLPGYFWYK_813.1_968.5
135
PSG9_HUMAN
LFIPQITR_494.3_614.4
136
количественн.
IS_LFIPQITR_499.3_624.4
136
PSG9_HUMAN
LFIPQITR_494.3_727.4
136
качественн.
IS_LFIPQITR_499.3_737.5
136
PTGDS_HUMAN
GPGEDFR_389.2_322.2
137
качественн.
IS_GPGEDFR_394.2_332.2
137
PTGDS_HUMAN
GPGEDFR_389.2_623.3
137
количественн.
IS_GPGEDFR_394.2_633.3
137
SHBG_HUMAN
ALALPPLGLAPLLNLWAKPQGR_770.5_256.2
27
количественн.
SHBG_HUMAN
ALALPPLGLAPLLNLWAKPQGR_770.5_457.3
27
качественн.
SHBG_HUMAN
IALGGLLFPASNLR_481.3_412.3
18
качественн.
IS_IALGGLLFPASNLR_484.6_412.3
18
SHBG_HUMAN
IALGGLLFPASNLR_481.3_657.4
18
количественн.
IS_IALGGLLFPASNLR_484.6_667.4
18
SOM2_HUMAN*
NYGLLYCFR_603.3_278.1
138
количественн.
IS_NYGLLYCFR_608.3_278.1
138
SOM2_HUMAN*
NYGLLYCFR_603.3_758.4
138
качественн.
IS_NYGLLYCFR_608.3_768.4
138
SOM2_HUMAN*
SVEGSCGF_421.7_223.1
139
качественн.
IS_SVEGSCGF_424.7_223.1
139
SOM2_HUMAN*
SVEGSCGF_421.7_383.1
139
количественн.
IS_SVEGSCGF_424.7_383.1
139
SPRL1_HUMAN
VLTHSELAPLR_412.6_512.3
140
количественн.
IS_VLTHSELAPLR_415.9_517.3
140
SPRL1_HUMAN
VLTHSELAPLR_412.6_568.8
140
качественн.
IS_VLTHSELAPLR_415.9_573.8
140
TENX_HUMAN
LNWEAPPGAFDSFLLR_917.0_414.2
141
качественн.
IS_LNWEAPPGAFDSFLLR_922.0_414.2
141
TENX_HUMAN
LNWEAPPGAFDSFLLR_917.0_614.3
141
количественн.
IS_LNWEAPPGAFDSFLLR_922.0_614.3
141
TENX_HUMAN
LSQLSVTDVTTSSLR_803.9_1165.6
142
количественн.
IS_LSQLSVTDVTTSSLR_808.9_1175.6
142
TENX_HUMAN
LSQLSVTDVTTSSLR_803.9_979.5
142
качественн.
IS_LSQLSVTDVTTSSLR_808.9_989.5
142
THBG_HUMAN
AVLHIGEK_289.5_292.2
143
качественн.
IS_AVLHIGEK_292.2_296.2
143
THBG_HUMAN
AVLHIGEK_289.5_348.7
143
количественн.
IS_AVLHIGEK_292.2_352.7
143
TIE1_HUMAN
VSWSLPLVPGPLVGDGFLLR_708.1_543.8
144
качественн.
IS_VSWSLPLVPGPLVGDGFLLR_711.4_548.8
144
TIE1_HUMAN
VSWSLPLVPGPLVGDGFLLR_708.1_620.9
144
количественн.
IS_VSWSLPLVPGPLVGDGFLLR_711.4_625.9
144
TRFE_HUMAN
YLGEEYVK_500.8_277.2
145
качественн.
Истощен
IS_YLGEEYVK_504.8_277.2
145
TRFE_HUMAN
YLGEEYVK_500.8_724.4
145
количественн.
Истощен
IS_YLGEEYVK_504.8_732.4
145
TTHY_HUMAN
VEIDTK_352.7_476.3
146
количественн.
Истощен
IS_VEIDTK_356.7_484.3
146
TTHY_HUMAN
VEIDTK_352.7_605.3
146
качественн.
Истощен
IS_VEIDTK_356.7_613.3
146
VTDB_HUMAN
ELPEHTVK_476.8_347.2
147
качественн.
IS_ELPEHTVK_480.8_355.2
147
VTDB_HUMAN
ELPEHTVK_476.8_710.4
147
количественн.
IS_ELPEHTVK_480.8_718.4
147
VTDB_HUMAN
VLEPTLK_400.3_458.3
148
качественн.
VTDB_HUMAN
VLEPTLK_400.3_587.3
148
количественн.
VTNC_HUMAN
GQYCYELDEK_652.8_1119.5
149
количественн.
IS_GQYCYELDEK_656.8_1127.5
149
VTNC_HUMAN
GQYCYELDEK_652.8_276.2
149
качественн.
IS_GQYCYELDEK_656.8_284.2
149
VTNC_HUMAN
VDTVDPPYPR_579.8_629.3
150
количественн.
IS_VDTVDPPYPR_584.8_639.3
150
VTNC_HUMAN
VDTVDPPYPR_579.8_744.4
150
качественн.
IS_VDTVDPPYPR_584.8_754.4
150
* Обозначает изменения названия. CSH обозначает, что пептид соответствует как CSH1, так и CSH2. HLAG здесь обозначают как HLACI, поскольку пептид превращается в несколько изотипов HLA I класса. LYAM3 здесь обозначают как LYAM1, поскольку, хотя пептидная последовательность присутствует в каждой, ее получают только посредством расщепления трипсином из LYAM1. SOM2 здесь обозначают как SOM2.CSH, поскольку пептиды специфичны как для SOM2, так и для CSH.
Таблица 22
Белок
Переход
SEQ ID №
119_13 2_aBM I_37
119_13 2_rBM I_37
119_13 2_aBM I_35
119_13 2_rBM I_35
126_13 9_aBM I_37
126_13 9_rBM I_37
126_13 9_aBM I_35
126_13 9_rBM I_35
133_14 6_aBM I_37
133_14 6_rBM I_37
A2GL
A2GL_DLLLPQPDLR
34
0,508
0,511
0,534
0,513
0,568
0,608
0,519
0,522
0,562
0,602
AFAM
AFAM_DADPDTFFAK
37
0,594
0,551
0,601
0,564
0,551
0,558
0,553
0,578
0,549
0,584
AFAM
AFAM_HFQNLGK
38
0,583
0,533
0,589
0,540
0,540
0,534
0,534
0,556
0,554
0,591
ALS
ALS_IRPHTFTGLSGLR
40
0,515
0,509
0,530
0,560
0,507
0,503
0,511
0,504
0,513
0,524
ANGT
ANGT_DPTFIPAPIQAK
42
0,577
0,622
0,666
0,685
0,574
0,608
0,573
0,616
0,507
0,544
APOC3
APOC3_GWVTDGFSSLK
47
0,582
0,576
0,510
0,548
0,605
0,611
0,607
0,593
0,635
0,617
APOH
APOH_ATVVYQGER
48
0,540
0,523
0,577
0,555
0,629
0,612
0,595
0,621
0,606
0,590
B2MG
B2MG_VEHSDLSFSK
50
0,533
0,566
0,502
0,507
0,522
0,505
0,512
0,521
0,558
0,548
B2MG
B2MG_VNHVTLSQPK
51
0,512
0,582
0,517
0,540
0,524
0,501
0,554
0,560
0,593
0,580
BGH3
BGH3_LTLLAPLNSVFK
52
0,562
0,575
0,557
0,610
0,509
0,512
0,531
0,538
0,522
0,557
C163A
C163A_INPASLDK
54
0,520
0,561
0,604
0,617
0,546
0,583
0,594
0,605
0,511
0,515
C1QB
C1QB_VPGLYYFTYHAS SR
55
0,573
0,607
0,538
0,573
0,586
0,631
0,565
0,560
0,596
0,588
CAH1
CAH1_GGPFSDSYR
56
0,560
0,560
0,563
0,553
0,500
0,590
0,563
0,605
0,543
0,581
CATD
CATD_VGFAEAAR
57
0,516
0,523
0,585
0,555
0,501
0,546
0,545
0,553
0,550
0,507
CATD
CATD_VSTLPAITLK
58
0,525
0,512
0,606
0,613
0,515
0,550
0,540
0,536
0,542
0,530
CBPN
CBPN_EALIQFLEQVHQG IK
59
0,508
0,563
0,567
0,585
0,508
0,509
0,549
0,582
0,517
0,507
CBPN
CBPN_NNANGVDLNR
60
0,514
0,519
0,579
0,606
0,521
0,507
0,502
0,516
0,505
0,526
CD14
CD14_LTVGAAQVPAQL LVGALR
61
0,532
0,539
0,578
0,626
0,608
0,623
0,635
0,665
0,605
0,577
CD14
CD14_SWLAELQQWLKP GLK
62
0,550
0,547
0,540
0,549
0,589
0,594
0,599
0,608
0,594
0,567
CFAB
CFAB_YGLVTYATYPK
64
0,635
0,605
0,514
0,522
0,574
0,556
0,530
0,572
0,564
0,592
CHL1
CHL1_VIAVNEVGR
66
0,670
0,680
0,669
0,661
0,550
0,592
0,549
0,554
0,514
0,522
CLUS
CLUS_ASSIIDELFQDR
67
0,563
0,557
0,713
0,667
0,554
0,530
0,685
0,649
0,526
0,541
CLUS
CLUS_LFDSDPITVTVPV EVSR
68
0,559
0,529
0,643
0,562
0,557
0,525
0,687
0,623
0,522
0,514
CO5
CO5_TLLPVSKPEIR
70
0,590
0,610
0,613
0,632
0,570
0,606
0,541
0,563
0,567
0,592
CO5
CO5_VFQFLEK
71
0,568
0,588
0,533
0,560
0,555
0,571
0,542
0,526
0,576
0,579
CO6
CO6_ALNHLPLEYNSAL YSR
72
0,619
0,647
0,721
0,795
0,553
0,537
0,592
0,621
0,524
0,541
CO8A
CO8A_SLLQPNK
74
0,512
0,536
0,515
0,537
0,574
0,566
0,561
0,547
0,610
0,638
CO8B
CO8B_QALEEFQK
76
0,528
0,522
0,522
0,500
0,576
0,563
0,546
0,501
0,605
0,633
CRIS3
CRIS3_AVSPPAR
78
0,513
0,543
0,592
0,626
0,508
0,562
0,567
0,587
0,530
0,577
CRIS3
CRIS3_YEDLYSNCK
79
0,530
0,539
0,631
0,654
0,529
0,574
0,596
0,616
0,544
0,602
CSH
CSH_AHQLAIDTYQEFEE TYIPK
80
0,615
0,550
0,534
0,516
0,568
0,566
0,570
0,540
0,527
0,540
CSH
CSH_ISLLLIESWLEPVR
81
0,597
0,528
0,515
0,506
0,530
0,521
0,550
0,520
0,543
0,558
ENPP2
ENPP2_TEFLSNYLTNVD DITLVPGTLGR
82
0,547
0,522
0,795
0,795
0,501
0,532
0,593
0,674
0,560
0,565
ENPP2
ENPP2_TYLHTYESEI
83
0,557
0,508
0,762
0,772
0,506
0,548
0,597
0,673
0,567
0,553
F13B
F13B_GDTYPAELYITGSI LR
84
0,563
0,557
0,528
0,533
0,569
0,576
0,588
0,623
0,503
0,516
FBLN1
FBLN1_TGYYFDGISR
86
0,609
0,585
0,506
0,560
0,594
0,581
0,562
0,519
0,536
0,550
FBLN3
FBLN3_IPSNPSHR
87
0,538
0,553
0,503
0,553
0,529
0,528
0,509
0,503
0,510
0,537
FETUA
FETUA_FSVVYAK
88
0,527
0,519
0,521
0,531
0,502
0,539
0,544
0,586
0,525
0,513
FETUA
FETUA_HTLNQIDEVK
89
0,532
0,506
0,559
0,599
0,503
0,528
0,587
0,618
0,523
0,514
HABP2
HABP2_FLNWIK
92
0,678
0,680
0,732
0,780
0,585
0,605
0,630
0,603
0,550
0,543
HEMO
HEMO_NFPSPVDAAFR
93
0,524
0,502
0,575
0,555
0,557
0,529
0,522
0,519
0,552
0,502
HLACI
HLACI_WAAVVVPSGEE QR
95
0,534
0,504
0,530
0,549
0,512
0,529
0,541
0,515
0,538
0,514
IBP1
IBP1_VVESLAK
97
0,578
0,556
0,559
0,516
0,546
0,528
0,557
0,542
0,516
0,518
IBP2
IBP2_LIQGAPTIR
98
0,539
0,525
0,521
0,518
0,505
0,507
0,517
0,563
0,501
0,500
IBP3
IBP3_FLNVLSPR
99
0,504
0,557
0,576
0,637
0,505
0,545
0,536
0,567
0,530
0,535
IBP3
IBP3_YGQPLPGYTTK
100
0,500
0,530
0,606
0,628
0,511
0,542
0,562
0,557
0,559
0,578
IBP4
IBP4_QCHPALDGQR
2
0,501
0,542
0,542
0,555
0,590
0,590
0,591
0,626
0,691
0,723
IBP6
IBP6_GAQTLYVPNCDHR
101
0,527
0,603
0,528
0,592
0,535
0,527
0,593
0,552
0,561
0,503
IBP6
IBP6_HLDSVLQQLQTEV YR
102
0,512
0,557
0,512
0,570
0,517
0,533
0,545
0,520
0,541
0,504
IGF2
IGF2_GIVEECCFR
103
0,523
0,545
0,704
0,743
0,542
0,558
0,627
0,667
0,567
0,573
INHBC
INHBC_LDFHFSSDR
107
0,575
0,534
0,573
0,643
0,586
0,595
0,530
0,522
0,619
0,665
ITIH3
ITIH3_ALDLSLK
111
0,525
0,534
0,522
0,564
0,529
0,527
0,516
0,518
0,544
0,519
ITIH4
ITIH4_ILDDLSPR
112
0,513
0,553
0,541
0,599
0,537
0,563
0,504
0,552
0,551
0,546
ITIH4
ITIH4_NPLVWVHASPEH VVVTR
113
0,501
0,532
0,586
0,595
0,573
0,589
0,505
0,514
0,587
0,556
ITIH4
ITIH4_QLGLPGPPDVPD HAAYHPF
114
0,518
0,506
0,537
0,565
0,547
0,538
0,523
0,568
0,526
0,520
KNG1
KNG1_DIPTNSPELEETLT HTITK
116
0,529
0,530
0,560
0,652
0,557
0,542
0,559
0,617
0,564
0,522
KNG1
KNG1_QVVAGLNFR
117
0,511
0,501
0,587
0,667
0,542
0,539
0,593
0,646
0,584
0,557
LBP
LBP_ITGFLKPGK
118
0,575
0,512
0,510
0,500
0,578
0,545
0,501
0,565
0,566
0,546
LBP
LBP_ITLPDFTGDLR
119
0,598
0,546
0,508
0,533
0,590
0,569
0,515
0,535
0,591
0,585
LYAM1
LYAM1_SYYWIGIR
120
0,550
0,600
0,574
0,654
0,567
0,623
0,666
0,720
0,542
0,580
NCAM1
NCAM1_GLGEISAASEFK
121
0,592
0,570
0,595
0,615
0,541
0,545
0,502
0,579
0,515
0,530
PAPP1
PAPP1_DIPHWLNPTR
122
0,571
0,505
0,531
0,524
0,505
0,520
0,525
0,569
0,533
0,512
PEDF
PEDF_LQSLFDSPDFSK
124
0,594
0,575
0,517
0,513
0,563
0,540
0,502
0,520
0,555
0,542
PEDF
PEDF_TVQAVLTVPK
125
0,604
0,607
0,574
0,603
0,584
0,598
0,561
0,601
0,560
0,557
PGRP2
PGRP2_AGLLRPDYALL GHR
126
0,581
0,575
0,590
0,577
0,539
0,583
0,528
0,592
0,507
0,527
PRDX2
PRDX2_GLFIIDGK
128
0,544
0,533
0,615
0,588
0,510
0,604
0,535
0,570
0,582
0,613
PRG2
PRG2_WNFAYWAAHQP WSR
129
0,564
0,527
0,507
0,538
0,545
0,507
0,515
0,511
0,590
0,535
PSG11
PSG11_LFIPQITPK
132
0,503
0,520
0,532
0,575
0,521
0,578
0,520
0,576
0,502
0,541
PSG1
PSG1_FQLPGQK
131
0,600
0,571
0,646
0,582
0,546
0,564
0,501
0,511
0,501
0,507
PSG2
PSG2_IHPSYTNYR
133
0,520
0,597
0,681
0,742
0,509
0,562
0,638
0,704
0,536
0,568
PSG3
PSG3_VSAPSGTGHLPGL NPL
134
0,650
0,577
0,568
0,533
0,604
0,603
0,549
0,546
0,606
0,634
PSG9
PSG9_DVLLLVHNLPQNL PGYFWYK
135
0,602
0,596
0,569
0,526
0,562
0,578
0,623
0,599
0,526
0,558
PSG9
PSG9_LFIPQITR
136
0,575
0,565
0,638
0,555
0,543
0,552
0,654
0,670
0,521
0,547
PTGDS
PTGDS_GPGEDFR
137
0,527
0,597
0,542
0,557
0,538
0,547
0,550
0,594
0,559
0,598
SHBG
SHBG_IALGGLLFPASNL R
18
0,580
0,567
0,563
0,641
0,575
0,574
0,562
0,532
0,588
0,586
SOM2. CSH
SOM2.CSH_NYGLLYCFR
138
0,604
0,535
0,547
0,509
0,531
0,515
0,525
0,517
0,554
0,590
SOM2. CSH
SOM2.CSH_SVEGSCGF
139
0,589
0,528
0,570
0,537
0,529
0,547
0,507
0,502
0,544
0,511
SPRL1
SPRL1_VLTHSELAPLR
140
0,534
0,545
0,528
0,526
0,516
0,583
0,533
0,504
0,530
0,557
TENX
TENX_LNWEAPPGAFDS FLLR
141
0,582
0,562
0,534
0,527
0,577
0,640
0,602
0,583
0,567
0,577
TENX
TENX_LSQLSVTDVTTSS LR
142
0,519
0,523
0,534
0,562
0,568
0,634
0,578
0,560
0,560
0,571
THBG
THBG_AVLHIGEK
143
0,558
0,571
0,503
0,564
0,506
0,521
0,509
0,507
0,550
0,554
TIE1
TIE1_VSWSLPLVPGPLV GDGFLLR
144
0,549
0,592
0,524
0,561
0,578
0,613
0,572
0,525
0,507
0,501
VTDB
VTDB_ELPEHTVK
147
0,557
0,538
0,516
0,586
0,506
0,515
0,517
0,530
0,529
0,512
VTNC
VTNC_GQYCYELDEK
149
0,663
0,674
0,641
0,694
0,612
0,638
0,618
0,641
0,582
0,606
VTNC
VTNC_VDTVDPPYPR
150
0,622
0,633
0,547
0,555
0,603
0,621
0,585
0,586
0,624
0,634
Таблица 23
Белок
Переход
SEQ ID №
133_14 6_aBM I_35
133_14 6_rBM I_35
140_15 3_aBM I_37
140_15 3_rBM I_37
140_15 3_aBM I_35
140_15 3_rBM I_35
119_15 3_aBM I_37
119_15 3_rBM I_37
119_15 3_aBM I_35
119_15 3_rBM I_35
A2GL
A2GL_DLLLPQPDLR
34
0,517
0,541
0,566
0,577
0,544
0,580
0,554
0,568
0,520
0,533
AFAM
AFAM_DADPDTFFAK
37
0,573
0,676
0,589
0,615
0,658
0,691
0,557
0,557
0,590
0,620
AFAM
AFAM_HFQNLGK
38
0,551
0,662
0,624
0,680
0,712
0,778
0,559
0,561
0,590
0,626
ALS
ALS_IRPHTFTGLSGLR
40
0,531
0,501
0,542
0,505
0,509
0,510
0,504
0,513
0,521
0,519
ANGT
ANGT_DPTFIPAPIQAK
42
0,567
0,547
0,557
0,601
0,555
0,586
0,549
0,589
0,558
0,593
APOC3
APOC3_GWVTDGFSSLK
47
0,672
0,596
0,674
0,635
0,591
0,501
0,622
0,599
0,587
0,539
APOH
APOH_ATVVYQGER
48
0,674
0,733
0,531
0,547
0,588
0,626
0,580
0,563
0,573
0,591
B2MG
B2MG_VEHSDLSFSK
50
0,519
0,591
0,599
0,596
0,553
0,503
0,543
0,520
0,536
0,508
B2MG
B2MG_VNHVTLSQPK
51
0,568
0,505
0,659
0,642
0,615
0,557
0,570
0,539
0,571
0,553
BGH3
BGH3_LTLLAPLNSVFK
52
0,562
0,621
0,595
0,623
0,532
0,553
0,542
0,564
0,516
0,518
C163A
C163A_INPASLDK
54
0,532
0,549
0,593
0,572
0,555
0,519
0,504
0,527
0,530
0,540
C1QB
C1QB_VPGLYYFTYHASSR
55
0,593
0,672
0,603
0,574
0,509
0,555
0,575
0,583
0,539
0,547
CAH1
CAH1_GGPFSDSYR
56
0,626
0,623
0,514
0,584
0,585
0,524
0,507
0,500
0,557
0,549
CATD
CATD_VGFAEAAR
57
0,660
0,637
0,693
0,686
0,819
0,817
0,576
0,547
0,626
0,634
CATD
CATD_VSTLPAITLK
58
0,674
0,719
0,671
0,692
0,802
0,866
0,564
0,549
0,623
0,651
CBPN
CBPN_EALIQFLEQVHQG IK
59
0,558
0,651
0,552
0,581
0,589
0,553
0,507
0,542
0,510
0,506
CBPN
CBPN_NNANGVDLNR
60
0,512
0,551
0,589
0,604
0,619
0,604
0,516
0,541
0,550
0,546
CD14
CD14_LTVGAAQVPAQL LVGALR
61
0,648
0,617
0,586
0,568
0,612
0,579
0,577
0,558
0,615
0,600
CD14
CD14_SWLAELQQWLK PGLK
62
0,635
0,583
0,585
0,560
0,638
0,579
0,577
0,554
0,601
0,569
CFAB
CFAB_YGLVTYATYPK
64
0,530
0,508
0,613
0,643
0,601
0,655
0,599
0,601
0,522
0,528
CHL1
CHL1_VIAVNEVGR
66
0,564
0,583
0,563
0,503
0,534
0,541
0,568
0,577
0,505
0,506
CLUS
CLUS_ASSIIDELFQDR
67
0,618
0,568
0,512
0,563
0,500
0,521
0,534
0,546
0,628
0,592
CLUS
CLUS_LFDSDPITVTVPV EVSR
68
0,669
0,615
0,512
0,517
0,562
0,611
0,521
0,511
0,610
0,543
CO5
CO5_TLLPVSKPEIR
70
0,528
0,540
0,580
0,560
0,531
0,566
0,577
0,589
0,548
0,525
CO5
CO5_VFQFLEK
71
0,547
0,522
0,599
0,568
0,556
0,515
0,577
0,575
0,540
0,511
CO6
CO6_ALNHLPLEYNSAL YSR
72
0,508
0,517
0,516
0,508
0,591
0,637
0,554
0,566
0,549
0,560
CO8A
CO8A_SLLQPNK
74
0,602
0,585
0,604
0,588
0,584
0,582
0,574
0,557
0,563
0,553
CO8B
CO8B_QALEEFQK
76
0,594
0,584
0,608
0,611
0,613
0,635
0,578
0,567
0,565
0,549
CRIS3
CRIS3_AVSPPAR
78
0,614
0,686
0,563
0,594
0,595
0,611
0,525
0,571
0,582
0,608
CRIS3
CRIS3_YEDLYSNCK
79
0,616
0,694
0,570
0,606
0,588
0,590
0,539
0,580
0,596
0,620
CSH
CSH_AHQLAIDTYQEFEE TYIPK
80
0,582
0,539
0,571
0,570
0,517
0,541
0,526
0,509
0,535
0,513
CSH
CSH_ISLLLIESWLEPVR
81
0,569
0,525
0,542
0,542
0,525
0,533
0,515
0,505
0,531
0,511
ENPP2
ENPP2_TEFLSNYLTNVD DITLVPGTLGR
82
0,583
0,509
0,703
0,737
0,650
0,659
0,574
0,571
0,517
0,558
ENPP2
ENPP2_TYLHTYESEI
83
0,591
0,519
0,711
0,722
0,692
0,664
0,576
0,559
0,500
0,545
F13B
F13B_GDTYPAELYITGS ILR
84
0,590
0,614
0,526
0,515
0,578
0,571
0,529
0,528
0,584
0,590
FBLN1
FBLN1_TGYYFDGISR
86
0,632
0,575
0,519
0,588
0,562
0,537
0,537
0,521
0,542
0,550
FBLN3
FBLN3_IPSNPSHR
87
0,575
0,670
0,528
0,524
0,617
0,649
0,523
0,506
0,545
0,559
FETUA
FETUA_FSVVYAK
88
0,568
0,692
0,658
0,652
0,659
0,660
0,552
0,534
0,530
0,517
FETUA
FETUA_HTLNQIDEVK
89
0,611
0,699
0,656
0,655
0,715
0,761
0,544
0,529
0,524
0,531
HABP2
HABP2_FLNWIK
92
0,605
0,517
0,623
0,587
0,516
0,541
0,595
0,592
0,591
0,544
HEMO
HEMO_NFPSPVDAAFR
93
0,509
0,587
0,634
0,658
0,502
0,514
0,567
0,536
0,501
0,538
HLACI
HLACI_WAAVVVPSGEE QR
95
0,537
0,538
0,574
0,591
0,604
0,552
0,527
0,543
0,502
0,508
IBP1
IBP1_VVESLAK
97
0,619
0,625
0,525
0,518
0,650
0,702
0,537
0,510
0,597
0,563
IBP2
IBP2_LIQGAPTIR
98
0,548
0,576
0,527
0,526
0,685
0,734
0,522
0,507
0,554
0,552
IBP3
IBP3_FLNVLSPR
99
0,514
0,532
0,525
0,574
0,581
0,611
0,532
0,566
0,507
0,514
IBP3
IBP3_YGQPLPGYTTK
100
0,525
0,525
0,548
0,617
0,505
0,521
0,547
0,581
0,552
0,540
IBP4
IBP4_QCHPALDGQR
2
0,600
0,624
0,677
0,688
0,530
0,568
0,608
0,606
0,539
0,530
IBP6
IBP6_GAQTLYVPNCD HR
101
0,569
0,557
0,501
0,524
0,593
0,562
0,522
0,533
0,526
0,505
IBP6
IBP6_HLDSVLQQLQTEV YR
102
0,519
0,539
0,521
0,514
0,559
0,523
0,520
0,521
0,511
0,506
IGF2
IGF2_GIVEECCFR
103
0,527
0,513
0,580
0,623
0,529
0,590
0,569
0,592
0,578
0,579
INHBC
INHBC_LDFHFSSDR
107
0,610
0,625
0,672
0,698
0,608
0,636
0,608
0,612
0,545
0,517
ITIH3
ITIH3_ALDLSLK
111
0,525
0,580
0,612
0,623
0,547
0,557
0,557
0,557
0,513
0,519
ITIH4
ITIH4_ILDDLSPR
112
0,500
0,546
0,538
0,510
0,521
0,626
0,531
0,537
0,501
0,508
ITIH4
ITIH4_NPLVWVHASPEH VVVTR
113
0,556
0,621
0,507
0,508
0,719
0,822
0,534
0,528
0,559
0,597
ITIH4
ITIH4_QLGLPGPPDVPD HAAYHPF
114
0,507
0,666
0,509
0,551
0,613
0,626
0,512
0,505
0,520
0,582
KNG1
KNG1_DIPTNSPELEETLT HTITK
116
0,522
0,599
0,609
0,593
0,512
0,588
0,558
0,535
0,523
0,525
KNG1
KNG1_QVVAGLNFR
117
0,581
0,528
0,664
0,653
0,634
0,548
0,562
0,554
0,602
0,602
LBP
LBP_ITGFLKPGK
118
0,521
0,593
0,640
0,645
0,542
0,615
0,587
0,559
0,505
0,565
LBP
LBP_ITLPDFTGDLR
119
0,524
0,581
0,672
0,681
0,505
0,550
0,609
0,588
0,524
0,521
LYAM1
LYAM1_SYYWIGIR
120
0,701
0,765
0,554
0,585
0,663
0,687
0,552
0,596
0,651
0,696
NCAM1
NCAM1_GLGEISAASEFK
121
0,587
0,501
0,515
0,505
0,535
0,553
0,536
0,531
0,501
0,557
PAPP1
PAPP1_DIPHWLNPTR
122
0,504
0,632
0,582
0,507
0,513
0,622
0,540
0,519
0,525
0,594
PEDF
PEDF_LQSLFDSPDFSK
124
0,560
0,580
0,616
0,659
0,615
0,649
0,577
0,571
0,545
0,565
PEDF
PEDF_TVQAVLTVPK
125
0,587
0,620
0,587
0,560
0,573
0,562
0,577
0,570
0,577
0,598
PGRP2
PGRP2_AGLLRPDYALL GHR
126
0,618
0,721
0,569
0,580
0,668
0,703
0,560
0,578
0,576
0,627
PRDX2
PRDX2_GLFIIDGK
128
0,620
0,598
0,551
0,537
0,609
0,542
0,525
0,530
0,547
0,536
PRG2
PRG2_WNFAYWAAHQP WSR
129
0,563
0,552
0,586
0,505
0,556
0,535
0,565
0,501
0,520
0,529
PSG11
PSG11_LFIPQITPK
132
0,570
0,523
0,562
0,544
0,517
0,604
0,511
0,516
0,500
0,501
PSG1
PSG1_FQLPGQK
131
0,606
0,524
0,558
0,577
0,510
0,548
0,524
0,509
0,508
0,535
PSG2
PSG2_IHPSYTNYR
133
0,592
0,721
0,551
0,544
0,515
0,537
0,522
0,563
0,597
0,660
PSG3
PSG3_VSAPSGTGHLPGL NPL
134
0,614
0,559
0,517
0,520
0,571
0,546
0,581
0,563
0,557
0,506
PSG9
PSG9_DVLLLVHNLPQNL PGYFWYK
135
0,535
0,516
0,525
0,604
0,605
0,669
0,546
0,567
0,547
0,500
PSG9
PSG9_LFIPQITR
136
0,527
0,541
0,506
0,585
0,573
0,617
0,528
0,541
0,571
0,554
PTGDS
PTGDS_GPGEDFR
137
0,603
0,671
0,567
0,595
0,535
0,521
0,536
0,532
0,537
0,545
SHBG
SHBG_IALGGLLFPASNL R
18
0,688
0,761
0,594
0,579
0,717
0,772
0,585
0,576
0,611
0,613
SOM2. CSH
SOM2.CSH_NYGLLYCFR
138
0,538
0,529
0,608
0,612
0,509
0,501
0,501
0,527
0,513
0,520
SOM2. CSH
SOM2.CSH_SVEGSCGF
139
0,538
0,579
0,531
0,539
0,612
0,684
0,525
0,539
0,542
0,565
SPRL1
SPRL1_VLTHSELAPLR
140
0,648
0,623
0,502
0,510
0,658
0,631
0,502
0,543
0,578
0,555
TENX
TENX_LNWEAPPGAFDS FLLR
141
0,622
0,569
0,614
0,530
0,531
0,682
0,576
0,573
0,550
0,519
TENX
TENX_LSQLSVTDVTTSS LR
142
0,606
0,546
0,571
0,519
0,644
0,825
0,550
0,542
0,508
0,588
THBG
THBG_AVLHIGEK
143
0,534
0,539
0,556
0,571
0,532
0,530
0,518
0,507
0,520
0,500
TIE1
TIE1_VSWSLPLVPGPLV GDGFLLR
144
0,551
0,526
0,578
0,559
0,527
0,515
0,513
0,531
0,536
0,502
VTDB
VTDB_ELPEHTVK
147
0,567
0,653
0,524
0,547
0,611
0,611
0,506
0,502
0,528
0,533
VTNC
VTNC_GQYCYELDEK
149
0,560
0,555
0,680
0,698
0,625
0,651
0,623
0,635
0,598
0,617
VTNC
VTNC_VDTVDPPYPR
150
0,595
0,566
0,673
0,670
0,568
0,533
0,625
0,629
0,574
0,553
Таблица 24
Белок
Переход
SEQ ID №
119_139 _aBMI _37
119_139 _rBMI _37
119_139 _aBMI _35
119_139 _rBMI _35
126_146 _aBMI _37
126_146 _rBMI _37
126_146 _aBMI _35
126_146 _rBMI _35
A2GL
A2GL_DLLLPQPDLR
34
0,549
0,565
0,503
0,500
0,552
0,596
0,511
0,513
AFAM
AFAM_DADPDTFFAK
37
0,543
0,531
0,554
0,579
0,575
0,594
0,586
0,633
AFAM
AFAM_HFQNLGK
38
0,526
0,501
0,529
0,544
0,579
0,593
0,568
0,612
ALS
ALS_IRPHTFTGLSGLR
40
0,516
0,526
0,532
0,533
0,536
0,530
0,518
0,511
ANGT
ANGT_DPTFIPAPIQAK
42
0,541
0,564
0,565
0,590
0,561
0,616
0,534
0,578
APOC3
APOC3_GWVTDGFSSLK
47
0,596
0,579
0,582
0,557
0,624
0,627
0,626
0,605
APOH
APOH_ATVVYQGER
48
0,604
0,573
0,567
0,577
0,597
0,591
0,610
0,654
B2MG
B2MG_VEHSDLSFSK
50
0,510
0,528
0,521
0,519
0,529
0,520
0,506
0,556
B2MG
B2MG_VNHVTLSQPK
51
0,523
0,531
0,545
0,538
0,556
0,539
0,563
0,535
BGH3
BGH3_LTLLAPLNSVFK
52
0,516
0,531
0,507
0,502
0,534
0,553
0,537
0,551
C163A
C163A_INPASLDK
54
0,532
0,578
0,573
0,581
0,511
0,534
0,568
0,596
C1QB
C1QB_VPGLYYFTYHASSR
55
0,564
0,582
0,544
0,544
0,606
0,640
0,556
0,572
CAH1
CAH1_GGPFSDSYR
56
0,500
0,550
0,547
0,569
0,502
0,546
0,571
0,578
CATD
CATD_VGFAEAAR
57
0,519
0,534
0,531
0,531
0,525
0,512
0,580
0,572
CATD
CATD_VSTLPAITLK
58
0,511
0,534
0,537
0,530
0,514
0,503
0,572
0,593
CBPN
CBPN_EALIQFLEQVHQGIK
59
0,514
0,519
0,527
0,544
0,501
0,518
0,528
0,580
CBPN
CBPN_NNANGVDLNR
60
0,525
0,504
0,510
0,506
0,506
0,530
0,530
0,504
CD14
CD14_LTVGAAQVPAQLLVGALR
61
0,573
0,558
0,616
0,623
0,604
0,611
0,647
0,662
CD14
CD14_SWLAELQQWLKPGLK
62
0,570
0,552
0,579
0,570
0,591
0,593
0,624
0,614
CFAB
CFAB_YGLVTYATYPK
64
0,590
0,573
0,520
0,548
0,582
0,591
0,510
0,501
CHL1
CHL1_VIAVNEVGR
66
0,570
0,610
0,518
0,516
0,563
0,578
0,541
0,556
CLUS
CLUS_ASSIIDELFQDR
67
0,545
0,533
0,695
0,666
0,547
0,548
0,644
0,590
CLUS
CLUS_LFDSDPITVTVPVEVSR
68
0,539
0,507
0,697
0,635
0,547
0,531
0,657
0,584
CO5
CO5_TLLPVSKPEIR
70
0,580
0,603
0,560
0,584
0,565
0,596
0,541
0,512
CO5
CO5_VFQFLEK
71
0,564
0,576
0,537
0,527
0,566
0,577
0,547
0,509
CO6
CO6_ALNHLPLEYNSALYSR
72
0,572
0,582
0,604
0,643
0,547
0,555
0,573
0,599
CO8A
CO8A_SLLQPNK
74
0,557
0,538
0,547
0,527
0,590
0,593
0,585
0,587
CO8B
CO8B_QALEEFQK
76
0,563
0,544
0,535
0,512
0,587
0,590
0,578
0,563
CRIS3
CRIS3_AVSPPAR
78
0,509
0,559
0,579
0,608
0,526
0,568
0,596
0,640
CRIS3
CRIS3_YEDLYSNCK
79
0,528
0,564
0,603
0,632
0,544
0,585
0,613
0,657
CSH
CSH_AHQLAIDTYQEFEETYIPK
80
0,574
0,571
0,555
0,535
0,519
0,505
0,574
0,559
CSH
CSH_ISLLLIESWLEPVR
81
0,543
0,531
0,535
0,517
0,501
0,514
0,561
0,544
ENPP2
ENPP2_TEFLSNYLTNVDDITLVPGTLGR
82
0,511
0,522
0,611
0,700
0,550
0,543
0,535
0,606
ENPP2
ENPP2_TYLHTYESEI
83
0,509
0,535
0,607
0,689
0,553
0,528
0,529
0,609
F13B
F13B_GDTYPAELYITGSILR
84
0,557
0,557
0,585
0,605
0,527
0,543
0,572
0,593
FBLN1
FBLN1_TGYYFDGISR
86
0,568
0,536
0,537
0,532
0,578
0,533
0,601
0,509
FBLN3
FBLN3_IPSNPSHR
87
0,522
0,503
0,503
0,505
0,527
0,524
0,550
0,571
FETUA
FETUA_FSVVYAK
88
0,504
0,544
0,533
0,575
0,531
0,509
0,543
0,610
FETUA
FETUA_HTLNQIDEVK
89
0,510
0,546
0,564
0,596
0,536
0,530
0,567
0,600
HABP2
HABP2_FLNWIK
92
0,576
0,593
0,630
0,598
0,606
0,610
0,650
0,615
HEMO
HEMO_NFPSPVDAAFR
93
0,534
0,530
0,505
0,545
0,559
0,547
0,505
0,557
HLACI
HLACI_WAAVVVPSGEEQR
95
0,503
0,513
0,556
0,542
0,503
0,521
0,525
0,525
IBP1
IBP1_VVESLAK
97
0,543
0,528
0,574
0,508
0,540
0,509
0,584
0,540
IBP2
IBP2_LIQGAPTIR
98
0,519
0,501
0,505
0,540
0,507
0,515
0,532
0,519
IBP3
IBP3_FLNVLSPR
99
0,537
0,565
0,556
0,588
0,506
0,531
0,504
0,518
IBP3
IBP3_YGQPLPGYTTK
100
0,544
0,563
0,583
0,582
0,508
0,542
0,538
0,518
IBP4
IBP4_QCHPALDGQR
2
0,572
0,552
0,573
0,589
0,626
0,636
0,563
0,568
IBP6
IBP6_GAQTLYVPNCDHR
101
0,532
0,540
0,584
0,531
0,528
0,531
0,544
0,515
IBP6
IBP6_HLDSVLQQLQTEVYR
102
0,519
0,543
0,546
0,504
0,516
0,522
0,508
0,501
IGF2
IGF2_GIVEECCFR
103
0,566
0,577
0,636
0,681
0,528
0,546
0,577
0,584
INHBC
INHBC_LDFHFSSDR
107
0,575
0,564
0,511
0,551
0,613
0,637
0,566
0,546
ITIH3
ITIH3_ALDLSLK
111
0,531
0,526
0,501
0,504
0,534
0,529
0,522
0,544
ITIH4
ITIH4_ILDDLSPR
112
0,528
0,544
0,512
0,558
0,541
0,567
0,507
0,506
ITIH4
ITIH4_NPLVWVHASPEHVVVTR
113
0,546
0,536
0,523
0,529
0,572
0,587
0,523
0,545
ITIH4
ITIH4_QLGLPGPPDVPDHAAYHPF
114
0,514
0,520
0,532
0,547
0,547
0,555
0,506
0,587
KNG1
KNG1_DIPTNSPELEETLTHTITK
116
0,535
0,503
0,537
0,577
0,568
0,565
0,535
0,549
KNG1
KNG1_QVVAGLNFR
117
0,517
0,506
0,590
0,628
0,571
0,570
0,594
0,607
LBP
LBP_ITGFLKPGK
118
0,562
0,511
0,523
0,526
0,582
0,562
0,507
0,591
LBP
LBP_ITLPDFTGDLR
119
0,576
0,533
0,536
0,500
0,601
0,595
0,506
0,565
LYAM1
LYAM1_SYYWIGIR
120
0,558
0,603
0,646
0,691
0,548
0,594
0,672
0,739
NCAM1
NCAM1_GLGEISAASEFK
121
0,567
0,556
0,524
0,559
0,527
0,535
0,505
0,565
PAPP1
PAPP1_DIPHWLNPTR
122
0,518
0,524
0,539
0,582
0,548
0,510
0,515
0,555
PEDF
PEDF_LQSLFDSPDFSK
124
0,558
0,530
0,514
0,526
0,577
0,570
0,527
0,540
PEDF
PEDF_TVQAVLTVPK
125
0,571
0,569
0,575
0,606
0,586
0,597
0,574
0,609
PGRP2
PGRP2_AGLLRPDYALLGHR
126
0,554
0,574
0,525
0,574
0,524
0,553
0,548
0,610
PRDX2
PRDX2_GLFIIDGK
128
0,511
0,567
0,517
0,531
0,533
0,577
0,551
0,548
PRG2
PRG2_WNFAYWAAHQPWSR
129
0,555
0,505
0,502
0,519
0,577
0,518
0,563
0,511
PSG11
PSG11_LFIPQITPK
132
0,505
0,552
0,508
0,565
0,515
0,556
0,542
0,558
PSG1
PSG1_FQLPGQK
131
0,560
0,570
0,502
0,508
0,529
0,531
0,522
0,505
PSG2
PSG2_IHPSYTNYR
133
0,511
0,570
0,656
0,714
0,528
0,572
0,609
0,713
PSG3
PSG3_VSAPSGTGHLPGLNPL
134
0,615
0,611
0,547
0,538
0,615
0,603
0,603
0,561
PSG9
PSG9_DVLLLVHNLPQNLPGYFWYK
135
0,557
0,555
0,625
0,597
0,560
0,595
0,546
0,507
PSG9
PSG9_LFIPQITR
136
0,539
0,529
0,649
0,656
0,547
0,576
0,565
0,547
PTGDS
PTGDS_GPGEDFR
137
0,520
0,502
0,542
0,572
0,540
0,551
0,561
0,594
SHBG
SHBG_IALGGLLFPASNLR
18
0,576
0,578
0,541
0,509
0,584
0,570
0,613
0,613
SOM2.CSH
SOM2.CSH_NYGLLYCFR
138
0,546
0,526
0,523
0,506
0,502
0,529
0,546
0,516
SOM2.CSH
SOM2.CSH_SVEGSCGF
139
0,538
0,551
0,503
0,510
0,502
0,525
0,551
0,583
SPRL1
SPRL1_VLTHSELAPLR
140
0,508
0,559
0,536
0,515
0,523
0,571
0,585
0,548
TENX
TENX_LNWEAPPGAFDSFLLR
141
0,556
0,590
0,590
0,568
0,588
0,606
0,598
0,536
TENX
TENX_LSQLSVTDVTTSSLR
142
0,538
0,573
0,562
0,544
0,567
0,591
0,566
0,505
THBG
THBG_AVLHIGEK
143
0,502
0,536
0,512
0,502
0,507
0,520
0,521
0,508
TIE1
TIE1_VSWSLPLVPGPLVGDGFLLR
144
0,559
0,596
0,563
0,514
0,537
0,550
0,553
0,506
VTDB
VTDB_ELPEHTVK
147
0,505
0,528
0,513
0,504
0,501
0,501
0,561
0,580
VTNC
VTNC_GQYCYELDEK
149
0,602
0,604
0,599
0,611
0,624
0,662
0,605
0,635
VTNC
VTNC_VDTVDPPYPR
150
0,601
0,599
0,582
0,573
0,627
0,651
0,582
0,570
Таблица 25
Белок
Переход
SEQ ID №
133_153 _aBMI _37
133_153 _rBMI _37
133_153 _aBMI _35
133_153 _rBMI _35
119_153 _aBMI _37
119_153 _rBMI _37
119_153 _aBMI _35
119_153 _rBMI _35
A2GL
A2GL_DLLLPQPDLR
34
0,581
0,597
0,538
0,549
0,554
0,568
0,520
0,533
AFAM
AFAM_DADPDTFFAK
37
0,539
0,561
0,584
0,647
0,557
0,557
0,590
0,620
AFAM
AFAM_HFQNLGK
38
0,549
0,584
0,595
0,679
0,559
0,561
0,590
0,626
ALS
ALS_IRPHTFTGLSGLR
40
0,503
0,515
0,517
0,503
0,504
0,513
0,521
0,519
ANGT
ANGT_DPTFIPAPIQAK
42
0,533
0,566
0,501
0,542
0,549
0,589
0,558
0,593
APOC3
APOC3_GWVTDGFSSLK
47
0,643
0,611
0,628
0,530
0,622
0,599
0,587
0,539
APOH
APOH_ATVVYQGER
48
0,601
0,584
0,632
0,656
0,580
0,563
0,573
0,591
B2MG
B2MG_VEHSDLSFSK
50
0,582
0,565
0,544
0,527
0,543
0,520
0,536
0,508
B2MG
B2MG_VNHVTLSQPK
51
0,613
0,599
0,582
0,551
0,570
0,539
0,571
0,553
BGH3
BGH3_LTLLAPLNSVFK
52
0,533
0,562
0,549
0,586
0,542
0,564
0,516
0,518
C163A
C163A_INPASLDK
54
0,524
0,505
0,509
0,515
0,504
0,527
0,530
0,540
C1QB
C1QB_VPGLYYFTYHASSR
55
0,579
0,564
0,577
0,613
0,575
0,583
0,539
0,547
CAH1
CAH1_GGPFSDSYR
56
0,522
0,531
0,610
0,598
0,507
0,500
0,557
0,549
CATD
CATD_VGFAEAAR
57
0,610
0,589
0,719
0,722
0,576
0,547
0,626
0,634
CATD
CATD_VSTLPAITLK
58
0,593
0,594
0,729
0,772
0,564
0,549
0,623
0,651
CBPN
CBPN_EALIQFLEQVHQGIK
59
0,504
0,530
0,516
0,552
0,507
0,542
0,510
0,506
CBPN
CBPN_NNANGVDLNR
60
0,529
0,549
0,533
0,510
0,516
0,541
0,550
0,546
CD14
CD14_LTVGAAQVPAQLLVGALR
61
0,600
0,567
0,620
0,582
0,577
0,558
0,615
0,600
CD14
CD14_SWLAELQQWLKPGLK
62
0,591
0,556
0,617
0,569
0,577
0,554
0,601
0,569
CFAB
CFAB_YGLVTYATYPK
64
0,579
0,596
0,529
0,523
0,599
0,601
0,522
0,528
CHL1
CHL1_VIAVNEVGR
66
0,515
0,519
0,564
0,573
0,568
0,577
0,505
0,506
CLUS
CLUS_ASSIIDELFQDR
67
0,518
0,539
0,591
0,560
0,534
0,546
0,628
0,592
CLUS
CLUS_LFDSDPITVTVPVEVSR
68
0,504
0,504
0,589
0,526
0,521
0,511
0,610
0,543
CO5
CO5_TLLPVSKPEIR
70
0,576
0,583
0,522
0,525
0,577
0,589
0,548
0,525
CO5
CO5_VFQFLEK
71
0,583
0,570
0,538
0,519
0,577
0,575
0,540
0,511
CO6
CO6_ALNHLPLEYNSALYSR
72
0,517
0,514
0,536
0,569
0,554
0,566
0,549
0,560
CO8A
CO8A_SLLQPNK
74
0,599
0,600
0,579
0,557
0,574
0,557
0,563
0,553
CO8B
CO8B_QALEEFQK
76
0,602
0,610
0,580
0,567
0,578
0,567
0,565
0,549
CRIS3
CRIS3_AVSPPAR
78
0,531
0,586
0,576
0,598
0,525
0,571
0,582
0,608
CRIS3
CRIS3_YEDLYSNCK
79
0,541
0,601
0,580
0,603
0,539
0,580
0,596
0,620
CSH
CSH_AHQLAIDTYQEFEETYIPK
80
0,523
0,533
0,550
0,513
0,526
0,509
0,535
0,513
CSH
CSH_ISLLLIESWLEPVR
81
0,531
0,546
0,548
0,511
0,515
0,505
0,531
0,511
ENPP2
ENPP2_TEFLSNYLTNVDDITLVPGT LGR
82
0,592
0,605
0,593
0,545
0,574
0,571
0,517
0,558
ENPP2
ENPP2_TYLHTYESEI
83
0,595
0,594
0,610
0,550
0,576
0,559
0,500
0,545
F13B
F13B_GDTYPAELYITGSILR
84
0,512
0,514
0,607
0,617
0,529
0,528
0,584
0,590
FBLN1
FBLN1_TGYYFDGISR
86
0,506
0,572
0,572
0,581
0,537
0,521
0,542
0,550
FBLN3
FBLN3_IPSNPSHR
87
0,515
0,524
0,571
0,622
0,523
0,506
0,545
0,559
FETUA
FETUA_FSVVYAK
88
0,562
0,558
0,532
0,508
0,552
0,534
0,530
0,517
FETUA
FETUA_HTLNQIDEVK
89
0,551
0,541
0,511
0,511
0,544
0,529
0,524
0,531
HABP2
HABP2_FLNWIK
92
0,556
0,546
0,539
0,562
0,595
0,592
0,591
0,544
HEMO
HEMO_NFPSPVDAAFR
93
0,588
0,552
0,526
0,528
0,567
0,536
0,501
0,538
HLACI
HLACI_WAAVVVPSGEEQR
95
0,522
0,564
0,518
0,513
0,527
0,543
0,502
0,508
IBP1
IBP1_VVESLAK
97
0,518
0,510
0,613
0,602
0,537
0,510
0,597
0,563
IBP2
IBP2_LIQGAPTIR
98
0,509
0,521
0,569
0,590
0,522
0,507
0,554
0,552
IBP3
IBP3_FLNVLSPR
99
0,545
0,568
0,525
0,541
0,532
0,566
0,507
0,514
IBP3
IBP3_YGQPLPGYTTK
100
0,573
0,608
0,529
0,501
0,547
0,581
0,552
0,540
IBP4
IBP4_QCHPALDGQR
2
0,674
0,694
0,571
0,570
0,608
0,606
0,539
0,530
IBP6
IBP6_GAQTLYVPNCDHR
101
0,549
0,502
0,541
0,529
0,522
0,533
0,526
0,505
IBP6
IBP6_HLDSVLQQLQTEVYR
102
0,540
0,501
0,515
0,524
0,520
0,521
0,511
0,506
IGF2
IGF2_GIVEECCFR
103
0,593
0,618
0,528
0,513
0,569
0,592
0,578
0,579
INHBC
INHBC_LDFHFSSDR
107
0,622
0,652
0,582
0,573
0,608
0,612
0,545
0,517
ITIH3
ITIH3_ALDLSLK
111
0,575
0,567
0,507
0,505
0,557
0,557
0,513
0,519
ITIH4
ITIH4_ILDDLSPR
112
0,542
0,524
0,518
0,559
0,531
0,537
0,501
0,508
ITIH4
ITIH4_NPLVWVHASPEHVVV TR
113
0,551
0,526
0,617
0,683
0,534
0,528
0,559
0,597
ITIH4
ITIH4_QLGLPGPPDVPDHAAY HPF
114
0,511
0,516
0,535
0,645
0,512
0,505
0,520
0,582
KNG1
KNG1_DIPTNSPELEETLTHT ITK
116
0,575
0,535
0,508
0,552
0,558
0,535
0,523
0,525
KNG1
KNG1_QVVAGLNFR
117
0,596
0,582
0,596
0,559
0,562
0,554
0,602
0,602
LBP
LBP_ITGFLKPGK
118
0,595
0,582
0,504
0,590
0,587
0,559
0,505
0,565
LBP
LBP_ITLPDFTGDLR
119
0,619
0,613
0,526
0,543
0,609
0,588
0,524
0,521
LYAM1
LYAM1_SYYWIGIR
120
0,557
0,603
0,678
0,708
0,552
0,596
0,651
0,696
NCAM1
NCAM1_GLGEISAASEFK
121
0,505
0,512
0,541
0,535
0,536
0,531
0,501
0,557
PAPP1
PAPP1_DIPHWLNPTR
122
0,516
0,542
0,530
0,640
0,540
0,519
0,525
0,594
PEDF
PEDF_LQSLFDSPDFSK
124
0,568
0,565
0,570
0,600
0,577
0,571
0,545
0,565
PEDF
PEDF_TVQAVLTVPK
125
0,566
0,552
0,574
0,592
0,577
0,570
0,577
0,598
PGRP2
PGRP2_AGLLRPDYALLGHR
126
0,549
0,578
0,645
0,717
0,560
0,578
0,576
0,627
PRDX2
PRDX2_GLFIIDGK
128
0,561
0,562
0,619
0,595
0,525
0,530
0,547
0,536
PRG2
PRG2_WNFAYWAAHQPWSR
129
0,564
0,504
0,522
0,569
0,565
0,501
0,520
0,529
PSG11
PSG11_LFIPQITPK
132
0,527
0,507
0,522
0,529
0,511
0,516
0,500
0,501
PSG1
PSG1_FQLPGQK
131
0,519
0,544
0,555
0,516
0,524
0,509
0,508
0,535
PSG2
PSG2_IHPSYTNYR
133
0,528
0,550
0,558
0,624
0,522
0,563
0,597
0,660
PSG3
PSG3_VSAPSGTGHLPGLNPL
134
0,546
0,558
0,550
0,508
0,581
0,563
0,557
0,506
PSG9
PSG9_DVLLLVHNLPQNLPGYF WYK
135
0,516
0,553
0,529
0,505
0,546
0,567
0,547
0,500
PSG9
PSG9_LFIPQITR
136
0,502
0,528
0,542
0,552
0,528
0,541
0,571
0,554
PTGDS
PTGDS_GPGEDFR
137
0,568
0,602
0,568
0,592
0,536
0,532
0,537
0,545
SHBG
SHBG_IALGGLLFPASNLR
18
0,588
0,585
0,686
0,728
0,585
0,576
0,611
0,613
SOM2.CSH
SOM2.CSH_NYGLLYCFR
138
0,560
0,588
0,511
0,546
0,501
0,527
0,513
0,520
SOM2.CSH
SOM2.CSH_SVEGSCGF
139
0,509
0,532
0,549
0,573
0,525
0,539
0,542
0,565
SPRL1
SPRL1_VLTHSELAPLR
140
0,512
0,533
0,619
0,582
0,502
0,543
0,578
0,555
TENX
TENX_LNWEAPPGAFDSFLLR
141
0,576
0,578
0,558
0,519
0,576
0,573
0,550
0,519
TENX
TENX_LSQLSVTDVTTSSLR
142
0,564
0,554
0,504
0,599
0,550
0,542
0,508
0,588
THBG
THBG_AVLHIGEK
143
0,557
0,547
0,527
0,529
0,518
0,507
0,520
0,500
TIE1
TIE1_VSWSLPLVPGPLVGDGF LLR
144
0,512
0,506
0,542
0,528
0,513
0,531
0,536
0,502
VTDB
VTDB_ELPEHTVK
147
0,542
0,524
0,551
0,597
0,506
0,502
0,528
0,533
VTNC
VTNC_GQYCYELDEK
149
0,603
0,618
0,573
0,581
0,623
0,635
0,598
0,617
VTNC
VTNC_VDTVDPPYPR
150
0,629
0,626
0,583
0,552
0,625
0,629
0,574
0,553
Таблица 26: белки под повышающей и понижающей регуляцией/переходы, используемые для обращений
Белок
Переход
SEQ ID №
Рег.
Белок
Переход
SEQ ID №
Рег.
AFAM
AFAM_DADPDTFFAK
37
повыш.
ITIH4
ITIH4_ILDDLSPR
112
пониж.
AFAM
AFAM_HFQNLGK
38
повыш.
PSG1
PSG1_FQLPGQK
131
пониж.
ANGT
ANGT_DPTFIPAPIQAK
42
повыш.
CHL1
CHL1_VIAVNEVGR
66
пониж.
APOC3
APOC3_GWVTDGFSSLK
47
повыш.
CSH
CSH_ISLLLIESWLEPVR
81
пониж.
APOH
APOH_ATVVYQGER
48
повыш.
NCAM1
NCAM1_GLGEISAASEFK
121
пониж.
CD14
CD14_LTVGAAQVPAQLLVGALR
61
повыш.
PRG2
PRG2_WNFAYWAAHQPWSR
129
пониж.
CD14
CD14_SWLAELQQWLKPGLK
62
повыш.
SOM2.CSH
SOM2.CSH_NYGLLYCFR
138
пониж.
CLUS
CLUS_ASSIIDELFQDR
67
повыш.
ALS
ALS_IRPHTFTGLSGLR
40
пониж.
CLUS
CLUS_LFDSDPITVTVPVEVSR
68
повыш.
FBLN1
FBLN1_TGYYFDGISR
86
пониж.
CO8A
CO8A_SLLQPNK
74
повыш.
PSG9
PSG9_LFIPQITR
136
пониж.
CO8B
CO8B_QALEEFQK
76
повыш.
CSH
CSH_AHQLAIDTYQEFEETYIPK
80
пониж.
F13B
F13B_GDTYPAELYITGSILR
84
повыш.
VTDB
VTDB_ELPEHTVK
147
пониж.
IBP4
IBP4_QCHPALDGQR
2
повыш.
IBP3
IBP3_FLNVLSPR
99
пониж.
PEDF
PEDF_LQSLFDSPDFSK
124
повыш.
IBP3
IBP3_YGQPLPGYTTK
100
пониж.
PEDF
PEDF_TVQAVLTVPK
125
повыш.
PSG9
PSG9_DVLLLVHNLPQNLPGYFWYK
135
пониж.
PRDX2
PRDX2_GLFIIDGK
128
повыш.
TENX
TENX_LSQLSVTDVTTSSLR
142
пониж.
PSG2
PSG2_IHPSYTNYR
133
повыш.
IBP1
IBP1_VVESLAK
97
пониж.
PTGDS
PTGDS_GPGEDFR
137
повыш.
IBP2
IBP2_LIQGAPTIR
98
пониж.
VTNC
VTNC_GQYCYELDEK
149
повыш.
SOM2.CSH
SOM2.CSH_SVEGSCGF
139
пониж.
VTNC
VTNC_VDTVDPPYPR
150
повыш.
SPRL1
SPRL1_VLTHSELAPLR
140
пониж.
B2MG
B2MG_VNHVTLSQPK
51
повыш.
TENX
TENX_LNWEAPPGAFDSFLLR
141
пониж.
BGH3
BGH3_LTLLAPLNSVFK
52
повыш.
TIE1
TIE1_VSWSLPLVPGPLVGDGFLLR
144
пониж.
CBPN
CBPN_NNANGVDLNR
60
повыш.
C163A
C163A_INPASLDK
54
пониж.
CO5
CO5_TLLPVSKPEIR
70
повыш.
IGF2
IGF2_GIVEECCFR
103
пониж.
CO5
CO5_VFQFLEK
71
повыш.
PSG3
PSG3_VSAPSGTGHLPGLNPL
134
пониж.
IBP6
IBP6_HLDSVLQQLQTEVYR
102
повыш.
CRIS3
CRIS3_AVSPPAR
78
пониж.
INHBC
INHBC_LDFHFSSDR
107
повыш.
CRIS3
CRIS3_YEDLYSNCK
79
пониж.
KNG1
KNG1_DIPTNSPELEETLTHTITK
116
повыш.
LYAM1
LYAM1_SYYWIGIR
120
пониж.
KNG1
KNG1_QVVAGLNFR
117
повыш.
PGRP2
PGRP2_AGLLRPDYALLGHR
126
пониж.
THBG
THBG_AVLHIGEK
143
повыш.
SHBG
SHBG_IALGGLLFPASNLR
18
пониж.
CATD
CATD_VGFAEAAR
57
повыш.
CO6
CO6_ALNHLPLEYNSALYSR
72
повыш.
ITIH4
ITIH4_NPLVWVHASPEHVVVTR
113
повыш.
A2GL
A2GL_DLLLPQPDLR
34
повыш.
CAH1
CAH1_GGPFSDSYR
56
повыш.
CATD
CATD_VSTLPAITLK
58
повыш.
CBPN
CBPN_EALIQFLEQVHQGIK
59
повыш.
HABP2
HABP2_FLNWIK
92
повыш.
CFAB
CFAB_YGLVTYATYPK
64
повыш.
HEMO
HEMO_NFPSPVDAAFR
93
повыш.
LBP
LBP_ITGFLKPGK
118
повыш.
LBP
LBP_ITLPDFTGDLR
119
повыш.
PAPP1
PAPP1_DIPHWLNPTR
122
повыш.
FETUA
FETUA_FSVVYAK
88
повыш.
FETUA
FETUA_HTLNQIDEVK
89
повыш.
IBP6
IBP6_GAQTLYVPNCDHR
101
повыш.
ITIH3
ITIH3_ALDLSLK
111
повыш.
B2MG
B2MG_VEHSDLSFSK
50
повыш.
ENPP2
ENPP2_TYLHTYESEI
83
повыш.
HLACI
HLACI_WAAVVVPSGEEQR
95
повыш.
ITIH4
ITIH4_QLGLPGPPDVPDHAAYHPF
114
повыш.
C1QB
C1QB_VPGLYYFTYHASSR
55
повыш.
ENPP2
ENPP2_TEFLSNYLTNVDDITLVPGTLGR
82
повыш.
FBLN3
FBLN3_IPSNPSHR
87
повыш.
PSG11
PSG11_LFIPQITPK
132
повыш.
Таблица 27: эффективность классификации по обращениям, недели 17 и 18.
AUROC обращения для недель беременности 17 и 18 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, без BMI-стратификации.
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
A2GL_DLLLPQPDLR_vs_CHL1_VIAVNEVGR
34 и 66
0,027
0,633
CO8B_QALEEFQK_vs_PSG3_VSAPSGTGHLPGLNPL
76 и 134
0,013
0,649
A2GL_DLLLPQPDLR_vs_PSG3_VSAPSGTGHLPGLNPL
34 и 134
0,036
0,626
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PSG3_VSAPSGTGHLPGLNPL
82 и 134
0,033
0,629
AFAM_DADPDTFFAK_vs_CHL1_VIAVNEVGR
37 и 66
0,005
0,669
ENPP2_TYLHTYESEI_vs_PSG3_VSAPSGTGHLPGLNPL
83 и 134
0,032
0,629
AFAM_DADPDTFFAK_vs_PSG3_VSAPSGTGHLPGLNPL
37 и 134
0,001
0,699
F13B_GDTYPAELYITGSILR_vs_CHL1_VIAVNEVGR
84 и 66
0,003
0,680
AFAM_DADPDTFFAK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
37 и 144
0,020
0,640
F13B_GDTYPAELYITGSILR_vs_CSH_AHQLAIDTYQEFEETYIPK
84 и 80
0,035
0,627
AFAM_DADPDTFFAK_vs_VTDB_ELPEHTVK
37 и 147
0,043
0,622
F13B_GDTYPAELYITGSILR_vs_FBLN1_TGYYFDGISR
84 и 86
0,035
0,627
AFAM_HFQNLGK_vs_CHL1_VIAVNEVGR
38 и 66
0,003
0,679
F13B_GDTYPAELYITGSILR_vs_PSG3_VSAPSGTGHLPGLNPL
84 и 134
0,007
0,663
AFAM_HFQNLGK_vs_NCAM1_GLGEISAASEFK
38 и 121
0,036
0,626
FBLN3_IPSNPSHR_vs_CHL1_VIAVNEVGR
87 и 66
0,049
0,619
AFAM_HFQNLGK_vs_PSG3_VSAPSGTGHLPGLNPL
38 и 134
0,004
0,676
FETUA_FSVVYAK_vs_CHL1_VIAVNEVGR
88 и 66
0,009
0,658
AFAM_HFQNLGK_vs_VTDB_ELPEHTVK
38 и 147
0,037
0,625
FETUA_FSVVYAK_vs_PGRP2_AGLLRPDYALLGHR
88 и 126
0,042
0,623
ANGT_DPTFIPAPIQAK_vs_CHL1_VIAVNEVGR
42 и 66
0,008
0,659
FETUA_FSVVYAK_vs_PSG3_VSAPSGTGHLPGLNPL
88 и 134
0,009
0,658
ANGT_DPTFIPAPIQAK_vs_CSH_AHQLAIDTYQEFEETYIPK
42 и 80
0,006
0,665
FETUA_HTLNQIDEVK_vs_CHL1_VIAVNEVGR
89 и 66
0,009
0,657
ANGT_DPTFIPAPIQAK_vs_CSH_ISLLLIESWLEPVR
42 и 81
0,023
0,637
FETUA_HTLNQIDEVK_vs_PSG3_VSAPSGTGHLPGLNPL
89 и 134
0,017
0,643
ANGT_DPTFIPAPIQAK_vs_FBLN1_TGYYFDGISR
42 и 86
0,025
0,635
HABP2_FLNWIK_vs_CHL1_VIAVNEVGR
92 и 66
0,000
0,739
ANGT_DPTFIPAPIQAK_vs_NCAM1_GLGEISAASEFK
42 и 121
0,040
0,624
HABP2_FLNWIK_vs_CSH_AHQLAIDTYQEFEETYIPK
92 и 80
0,002
0,687
ANGT_DPTFIPAPIQAK_vs_PSG1_FQLPGQK
42 и 131
0,036
0,626
HABP2_FLNWIK_vs_CSH_ISLLLIESWLEPVR
92 и 81
0,005
0,667
ANGT_DPTFIPAPIQAK_vs_PSG3_VSAPSGTGHLPGLNPL
42 и 134
0,004
0,675
HABP2_FLNWIK_vs_FBLN1_TGYYFDGISR
92 и 86
0,003
0,679
ANGT_DPTFIPAPIQAK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
42 и 135
0,042
0,623
HABP2_FLNWIK_vs_IBP3_FLNVLSPR
92 и 99
0,038
0,625
ANGT_DPTFIPAPIQAK_vs_SOM2.CSH_NYGLLYCFR
42 и 138
0,012
0,651
HABP2_FLNWIK_vs_IBP3_YGQPLPGYTTK
92 и 100
0,029
0,631
ANGT_DPTFIPAPIQAK_vs_SOM2.CSH_SVEGSCGF
42 и 139
0,032
0,631
HABP2_FLNWIK_vs_IGF2_GIVEECCFR
92 и 103
0,008
0,659
APOC3_GWVTDGFSSLK_vs_CHL1_VIAVNEVGR
47 и 66
0,047
0,619
HABP2_FLNWIK_vs_ITIH4_ILDDLSPR
92 и 112
0,003
0,680
APOC3_GWVTDGFSSLK_vs_PSG3_VSAPSGTGHLPGLNPL
47 и 134
0,021
0,639
HABP2_FLNWIK_vs_LYAM1_SYYWIGIR
92 и 120
0,018
0,643
APOH_ATVVYQGER_vs_CHL1_VIAVNEVGR
48 и 66
0,008
0,660
HABP2_FLNWIK_vs_NCAM1_GLGEISAASEFK
92 и 121
0,002
0,683
APOH_ATVVYQGER_vs_CSH_AHQLAIDTYQEFEETYIPK
48 и 80
0,039
0,624
HABP2_FLNWIK_vs_PGRP2_AGLLRPDYALLGHR
92 и 126
0,004
0,672
APOH_ATVVYQGER_vs_PSG3_VSAPSGTGHLPGLNPL
48 и 134
0,024
0,636
HABP2_FLNWIK_vs_PRG2_WNFAYWAAHQPWSR
92 и 129
0,027
0,633
B2MG_VEHSDLSFSK_vs_CHL1_VIAVNEVGR
50 и 66
0,042
0,622
HABP2_FLNWIK_vs_PSG1_FQLPGQK
92 и 131
0,014
0,649
B2MG_VNHVTLSQPK_vs_CHL1_VIAVNEVGR
51 и 66
0,023
0,637
HABP2_FLNWIK_vs_PSG3_VSAPSGTGHLPGLNPL
92 и 134
0,000
0,750
B2MG_VNHVTLSQPK_vs_PSG3_VSAPSGTGHLPGLNPL
51 и 134
0,046
0,620
HABP2_FLNWIK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
92 и 135
0,015
0,646
BGH3_LTLLAPLNSVFK_vs_CHL1_VIAVNEVGR
52 и 66
0,006
0,666
HABP2_FLNWIK_vs_PSG9_LFIPQITR
92 и 136
0,043
0,622
BGH3_LTLLAPLNSVFK_vs_CSH_AHQLAIDTYQEFEETYIPK
52 и 80
0,014
0,648
HABP2_FLNWIK_vs_SHBG_IALGGLLFPASNLR
92 и 18
0,010
0,655
BGH3_LTLLAPLNSVFK_vs_CSH_ISLLLIESWLEPVR
52 и 81
0,050
0,618
HABP2_FLNWIK_vs_SOM2.CSH_NYGLLYCFR
92 и 138
0,004
0,674
BGH3_LTLLAPLNSVFK_vs_FBLN1_TGYYFDGISR
52 и 86
0,020
0,640
HABP2_FLNWIK_vs_SOM2.CSH_SVEGSCGF
92 и 139
0,005
0,673
BGH3_LTLLAPLNSVFK_vs_PSG3_VSAPSGTGHLPGLNPL
52 и 134
0,024
0,636
HABP2_FLNWIK_vs_TENX_LNWEAPPGAFDSFLLR
92 и 141
0,016
0,645
BGH3_LTLLAPLNSVFK_vs_SHBG_IALGGLLFPASNLR
52 и 18
0,029
0,632
HABP2_FLNWIK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
92 и 144
0,004
0,673
BGH3_LTLLAPLNSVFK_vs_SOM2.CSH_NYGLLYCFR
52 и 138
0,043
0,622
HABP2_FLNWIK_vs_VTDB_ELPEHTVK
92 и 147
0,000
0,711
C1QB_VPGLYYFTYHASSR_vs_CHL1_VIAVNEVGR
55 и 66
0,018
0,643
HEMO_NFPSPVDAAFR_vs_CHL1_VIAVNEVGR
93 и 66
0,004
0,673
C1QB_VPGLYYFTYHASSR_vs_CSH_AHQLAIDTYQEFEETYIPK
55 и 80
0,010
0,655
HEMO_NFPSPVDAAFR_vs_CSH_AHQLAIDTYQEFEETYIPK
93 и 80
0,047
0,619
C1QB_VPGLYYFTYHASSR_vs_CSH_ISLLLIESWLEPVR
55 и 81
0,016
0,645
HEMO_NFPSPVDAAFR_vs_PSG3_VSAPSGTGHLPGLNPL
93 и 134
0,016
0,645
C1QB_VPGLYYFTYHASSR_vs_FBLN1_TGYYFDGISR
55 и 86
0,046
0,620
IBP4_QCHPALDGQR_vs_CHL1_VIAVNEVGR
2 и 66
0,023
0,637
C1QB_VPGLYYFTYHASSR_vs_IBP2_LIQGAPTIR
55 и 98
0,017
0,644
IBP4_QCHPALDGQR_vs_PSG3_VSAPSGTGHLPGLNPL
2 и 134
0,021
0,640
C1QB_VPGLYYFTYHASSR_vs_PGRP2_AGLLRPDYALLGHR
55 и 126
0,032
0,629
IBP6_HLDSVLQQLQTEVYR_vs_PSG3_VSAPSGTGHLPGLNPL
102 и 134
0,032
0,629
C1QB_VPGLYYFTYHASSR_vs_PSG3_VSAPSGTGHLPGLNPL
55 и 134
0,028
0,632
INHBC_LDFHFSSDR_vs_CHL1_VIAVNEVGR
107 и 66
0,010
0,655
C1QB_VPGLYYFTYHASSR_vs_SHBG_IALGGLLFPASNLR
55 и 18
0,036
0,627
INHBC_LDFHFSSDR_vs_CSH_AHQLAIDTYQEFEETYIPK
107 и 80
0,050
0,618
C1QB_VPGLYYFTYHASSR_vs_SOM2.CSH_NYGLLYCFR
55 и 138
0,011
0,653
INHBC_LDFHFSSDR_vs_PRG2_WNFAYWAAHQPWSR
107 и 129
0,048
0,619
C1QB_VPGLYYFTYHASSR_vs_SOM2.CSH_SVEGSCGF
55 и 139
0,048
0,621
INHBC_LDFHFSSDR_vs_PSG3_VSAPSGTGHLPGLNPL
107 и 134
0,007
0,663
CBPN_EALIQFLEQVHQGIK_vs_PSG3_VSAPSGTGHLPGLNPL
59 и 134
0,038
0,625
INHBC_LDFHFSSDR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
107 и 135
0,049
0,619
CBPN_EALIQFLEQVHQGIK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
59 и 135
0,037
0,626
ITIH3_ALDLSLK_vs_PSG3_VSAPSGTGHLPGLNPL
111 и 134
0,041
0,623
CBPN_NNANGVDLNR_vs_CHL1_VIAVNEVGR
60 и 66
0,044
0,621
KNG1_DIPTNSPELEETLTHTITK_vs_CHL1_VIAVNEVGR
116 и 66
0,013
0,650
CBPN_NNANGVDLNR_vs_PSG3_VSAPSGTGHLPGLNPL
60 и 134
0,044
0,622
KNG1_DIPTNSPELEETLTHTITK_vs_CSH_AHQLAIDTYQEFEETYIPK
116 и 80
0,032
0,629
CD14_LTVGAAQVPAQLLVGALR_vs_CHL1_VIAVNEVGR
61 и 66
0,011
0,652
KNG1_DIPTNSPELEETLTHTITK_vs_PSG3_VSAPSGTGHLPGLNPL
116 и 134
0,007
0,661
CD14_LTVGAAQVPAQLLVGALR_vs_CSH_AHQLAIDTYQEFEETYIPK
61 и 80
0,046
0,620
KNG1_DIPTNSPELEETLTHTITK_vs_SOM2.CSH_NYGLLYCFR
116 и 138
0,049
0,619
CD14_LTVGAAQVPAQLLVGALR_vs_PSG3_VSAPSGTGHLPGLNPL
61 и 134
0,032
0,629
KNG1_QVVAGLNFR_vs_CHL1_VIAVNEVGR
117 и 66
0,023
0,637
CD14_SWLAELQQWLKPGLK_vs_CHL1_VIAVNEVGR
62 и 66
0,005
0,667
KNG1_QVVAGLNFR_vs_PSG3_VSAPSGTGHLPGLNPL
117 и 134
0,041
0,623
CD14_SWLAELQQWLKPGLK_vs_CSH_AHQLAIDTYQEFEETYIPK
62 и 80
0,024
0,636
LBP_ITGFLKPGK_vs_CHL1_VIAVNEVGR
118 и 66
0,024
0,636
CD14_SWLAELQQWLKPGLK_vs_PSG3_VSAPSGTGHLPGLNPL
62 и 134
0,015
0,646
LBP_ITGFLKPGK_vs_PSG3_VSAPSGTGHLPGLNPL
118 и 134
0,011
0,654
CFAB_YGLVTYATYPK_vs_C163A_INPASLDK
64 и 54
0,045
0,621
LBP_ITLPDFTGDLR_vs_CHL1_VIAVNEVGR
119 и 66
0,008
0,661
CFAB_YGLVTYATYPK_vs_CHL1_VIAVNEVGR
64 и 66
0,001
0,703
LBP_ITLPDFTGDLR_vs_CSH_AHQLAIDTYQEFEETYIPK
119 и 80
0,041
0,623
CFAB_YGLVTYATYPK_vs_CSH_AHQLAIDTYQEFEETYIPK
64 и 80
0,011
0,654
LBP_ITLPDFTGDLR_vs_FBLN1_TGYYFDGISR
119 и 86
0,049
0,619
CFAB_YGLVTYATYPK_vs_CSH_ISLLLIESWLEPVR
64 и 81
0,024
0,636
LBP_ITLPDFTGDLR_vs_LYAM1_SYYWIGIR
119 и 120
0,046
0,620
CFAB_YGLVTYATYPK_vs_FBLN1_TGYYFDGISR
64 и 86
0,023
0,637
LBP_ITLPDFTGDLR_vs_NCAM1_GLGEISAASEFK
119 и 121
0,042
0,623
CFAB_YGLVTYATYPK_vs_LYAM1_SYYWIGIR
64 и 120
0,034
0,628
LBP_ITLPDFTGDLR_vs_PSG3_VSAPSGTGHLPGLNPL
119 и 134
0,006
0,665
CFAB_YGLVTYATYPK_vs_NCAM1_GLGEISAASEFK
64 и 121
0,005
0,671
LBP_ITLPDFTGDLR_vs_SHBG_IALGGLLFPASNLR
119 и 18
0,047
0,619
CFAB_YGLVTYATYPK_vs_PGRP2_AGLLRPDYALLGHR
64 и 126
0,012
0,651
LBP_ITLPDFTGDLR_vs_SOM2.CSH_SVEGSCGF
119 и 139
0,049
0,620
CFAB_YGLVTYATYPK_vs_PSG1_FQLPGQK
64 и 131
0,032
0,629
LBP_ITLPDFTGDLR_vs_VTDB_ELPEHTVK
119 и 147
0,019
0,641
CFAB_YGLVTYATYPK_vs_PSG3_VSAPSGTGHLPGLNPL
64 и 134
0,000
0,722
PEDF_LQSLFDSPDFSK_vs_CHL1_VIAVNEVGR
124 и 66
0,003
0,681
CFAB_YGLVTYATYPK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
64 и 135
0,031
0,630
PEDF_LQSLFDSPDFSK_vs_CSH_AHQLAIDTYQEFEETYIPK
124 и 80
0,020
0,640
CFAB_YGLVTYATYPK_vs_SHBG_IALGGLLFPASNLR
64 и 18
0,015
0,646
PEDF_LQSLFDSPDFSK_vs_CSH_ISLLLIESWLEPVR
124 и 81
0,044
0,621
CFAB_YGLVTYATYPK_vs_SOM2.CSH_NYGLLYCFR
64 и 138
0,020
0,640
PEDF_LQSLFDSPDFSK_vs_FBLN1_TGYYFDGISR
124 и 86
0,026
0,634
CFAB_YGLVTYATYPK_vs_SOM2.CSH_SVEGSCGF
64 и 139
0,027
0,635
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,050
0,618
CFAB_YGLVTYATYPK_vs_TENX_LNWEAPPGAFDSFLLR
64 и 141
0,031
0,630
PEDF_LQSLFDSPDFSK_vs_PSG1_FQLPGQK
124 и 131
0,025
0,635
CFAB_YGLVTYATYPK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
64 и 144
0,012
0,652
PEDF_LQSLFDSPDFSK_vs_PSG3_VSAPSGTGHLPGLNPL
124 и 134
0,006
0,666
CFAB_YGLVTYATYPK_vs_VTDB_ELPEHTVK
64 и 147
0,006
0,664
PEDF_LQSLFDSPDFSK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
124 и 135
0,036
0,627
CLUS_ASSIIDELFQDR_vs_CHL1_VIAVNEVGR
67 и 66
0,002
0,682
PEDF_LQSLFDSPDFSK_vs_SOM2.CSH_NYGLLYCFR
124 и 138
0,027
0,633
CLUS_ASSIIDELFQDR_vs_CSH_AHQLAIDTYQEFEETYIPK
67 и 80
0,027
0,633
PEDF_LQSLFDSPDFSK_vs_SOM2.CSH_SVEGSCGF
124 и 139
0,049
0,620
CLUS_ASSIIDELFQDR_vs_FBLN1_TGYYFDGISR
67 и 86
0,028
0,633
PEDF_LQSLFDSPDFSK_vs_TENX_LNWEAPPGAFDSFLLR
124 и 141
0,043
0,622
CLUS_ASSIIDELFQDR_vs_NCAM1_GLGEISAASEFK
67 и 121
0,042
0,622
PEDF_LQSLFDSPDFSK_vs_VTDB_ELPEHTVK
124 и 147
0,025
0,635
CLUS_ASSIIDELFQDR_vs_PSG3_VSAPSGTGHLPGLNPL
67 и 134
0,007
0,664
PEDF_TVQAVLTVPK_vs_CHL1_VIAVNEVGR
125 и 66
0,004
0,675
CLUS_ASSIIDELFQDR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
67 и 135
0,035
0,627
PEDF_TVQAVLTVPK_vs_CSH_AHQLAIDTYQEFEETYIPK
125 и 80
0,015
0,646
CLUS_ASSIIDELFQDR_vs_VTDB_ELPEHTVK
67 и 147
0,032
0,629
PEDF_TVQAVLTVPK_vs_CSH_ISLLLIESWLEPVR
125 и 81
0,039
0,624
CLUS_LFDSDPITVTVPVEVSR_vs_CHL1_VIAVNEVGR
68 и 66
0,003
0,679
PEDF_TVQAVLTVPK_vs_FBLN1_TGYYFDGISR
125 и 86
0,018
0,642
CLUS_LFDSDPITVTVPVEVSR_vs_CSH_AHQLAIDTYQEFEETYIPK
68 и 80
0,047
0,620
PEDF_TVQAVLTVPK_vs_NCAM1_GLGEISAASEFK
125 и 121
0,033
0,628
CLUS_LFDSDPITVTVPVEVSR_vs_FBLN1_TGYYFDGISR
68 и 86
0,028
0,633
PEDF_TVQAVLTVPK_vs_PSG1_FQLPGQK
125 и 131
0,032
0,629
CLUS_LFDSDPITVTVPVEVSR_vs_PSG3_VSAPSGTGHLPGLNPL
68 и 134
0,014
0,648
PEDF_TVQAVLTVPK_vs_PSG3_VSAPSGTGHLPGLNPL
125 и 134
0,004
0,673
CLUS_LFDSDPITVTVPVEVSR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
68 и 135
0,034
0,628
PEDF_TVQAVLTVPK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
125 и 135
0,050
0,618
CLUS_LFDSDPITVTVPVEVSR_vs_VTDB_ELPEHTVK
68 и 147
0,038
0,625
PEDF_TVQAVLTVPK_vs_SOM2.CSH_NYGLLYCFR
125 и 138
0,040
0,624
CO5_TLLPVSKPEIR_vs_CHL1_VIAVNEVGR
70 и 66
0,002
0,683
PEDF_TVQAVLTVPK_vs_VTDB_ELPEHTVK
125 и 147
0,023
0,637
CO5_TLLPVSKPEIR_vs_CSH_AHQLAIDTYQEFEETYIPK
70 и 80
0,021
0,640
VTNC_GQYCYELDEK_vs_CHL1_VIAVNEVGR
149 и 66
0,000
0,719
CO5_TLLPVSKPEIR_vs_FBLN1_TGYYFDGISR
70 и 86
0,033
0,628
VTNC_GQYCYELDEK_vs_CSH_AHQLAIDTYQEFEETYIPK
149 и 80
0,006
0,665
CO5_TLLPVSKPEIR_vs_NCAM1_GLGEISAASEFK
70 и 121
0,033
0,628
VTNC_GQYCYELDEK_vs_CSH_ISLLLIESWLEPVR
149 и 81
0,013
0,650
CO5_TLLPVSKPEIR_vs_PGRP2_AGLLRPDYALLGHR
70 и 126
0,043
0,622
VTNC_GQYCYELDEK_vs_FBLN1_TGYYFDGISR
149 и 86
0,008
0,659
CO5_TLLPVSKPEIR_vs_PSG3_VSAPSGTGHLPGLNPL
70 и 134
0,002
0,688
VTNC_GQYCYELDEK_vs_IGF2_GIVEECCFR
149 и 103
0,046
0,620
CO5_TLLPVSKPEIR_vs_SOM2.CSH_NYGLLYCFR
70 и 138
0,038
0,625
VTNC_GQYCYELDEK_vs_ITIH4_ILDDLSPR
149 и 112
0,040
0,624
CO5_VFQFLEK_vs_CHL1_VIAVNEVGR
71 и 66
0,002
0,686
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,020
0,640
CO5_VFQFLEK_vs_CSH_AHQLAIDTYQEFEETYIPK
71 и 80
0,020
0,640
VTNC_GQYCYELDEK_vs_NCAM1_GLGEISAASEFK
149 и 121
0,008
0,659
CO5_VFQFLEK_vs_FBLN1_TGYYFDGISR
71 и 86
0,033
0,628
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,005
0,670
CO5_VFQFLEK_vs_NCAM1_GLGEISAASEFK
71 и 121
0,034
0,628
VTNC_GQYCYELDEK_vs_PSG1_FQLPGQK
149 и 131
0,021
0,639
CO5_VFQFLEK_vs_PGRP2_AGLLRPDYALLGHR
71 и 126
0,046
0,620
VTNC_GQYCYELDEK_vs_PSG3_VSAPSGTGHLPGLNPL
149 и 134
0,000
0,743
CO5_VFQFLEK_vs_PSG3_VSAPSGTGHLPGLNPL
71 и 134
0,002
0,685
VTNC_GQYCYELDEK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
149 и 135
0,029
0,631
CO5_VFQFLEK_vs_SHBG_IALGGLLFPASNLR
71 и 18
0,043
0,622
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,014
0,648
CO5_VFQFLEK_vs_SOM2.CSH_NYGLLYCFR
71 и 138
0,038
0,625
VTNC_GQYCYELDEK_vs_SOM2.CSH_NYGLLYCFR
149 и 138
0,010
0,655
CO5_VFQFLEK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
71 и 144
0,047
0,619
VTNC_GQYCYELDEK_vs_SOM2.CSH_SVEGSCGF
149 и 139
0,009
0,660
CO6_ALNHLPLEYNSALYSR_vs_CHL1_VIAVNEVGR
72 и 66
0,000
0,723
VTNC_GQYCYELDEK_vs_TENX_LNWEAPPGAFDSFLLR
149 и 141
0,023
0,637
CO6_ALNHLPLEYNSALYSR_vs_CSH_AHQLAIDTYQEFEETYIPK
72 и 80
0,010
0,655
VTNC_GQYCYELDEK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
149 и 144
0,006
0,665
CO6_ALNHLPLEYNSALYSR_vs_CSH_ISLLLIESWLEPVR
72 и 81
0,035
0,627
VTNC_GQYCYELDEK_vs_VTDB_ELPEHTVK
149 и 147
0,001
0,694
CO6_ALNHLPLEYNSALYSR_vs_FBLN1_TGYYFDGISR
72 и 86
0,013
0,649
VTNC_VDTVDPPYPR_vs_CHL1_VIAVNEVGR
150 и 66
0,000
0,712
CO6_ALNHLPLEYNSALYSR_vs_NCAM1_GLGEISAASEFK
72 и 121
0,010
0,656
VTNC_VDTVDPPYPR_vs_CSH_AHQLAIDTYQEFEETYIPK
150 и 80
0,010
0,654
CO6_ALNHLPLEYNSALYSR_vs_PGRP2_AGLLRPDYALLGHR
72 и 126
0,049
0,619
VTNC_VDTVDPPYPR_vs_CSH_ISLLLIESWLEPVR
150 и 81
0,029
0,631
CO6_ALNHLPLEYNSALYSR_vs_PSG1_FQLPGQK
72 и 131
0,043
0,622
VTNC_VDTVDPPYPR_vs_FBLN1_TGYYFDGISR
150 и 86
0,034
0,628
CO6_ALNHLPLEYNSALYSR_vs_PSG3_VSAPSGTGHLPGLNPL
72 и 134
0,001
0,699
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,034
0,628
CO6_ALNHLPLEYNSALYSR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
72 и 135
0,041
0,623
VTNC_VDTVDPPYPR_vs_NCAM1_GLGEISAASEFK
150 и 121
0,021
0,640
CO6_ALNHLPLEYNSALYSR_vs_SOM2.CSH_NYGLLYCFR
72 и 138
0,019
0,641
VTNC_VDTVDPPYPR_vs_PGRP2_AGLLRPDYALLGHR
150 и 126
0,039
0,625
CO6_ALNHLPLEYNSALYSR_vs_SOM2.CSH_SVEGSCGF
72 и 139
0,028
0,634
VTNC_VDTVDPPYPR_vs_PSG1_FQLPGQK
150 и 131
0,033
0,628
CO6_ALNHLPLEYNSALYSR_vs_TENX_LNWEAPPGAFDSFLLR
72 и 141
0,036
0,626
VTNC_VDTVDPPYPR_vs_PSG3_VSAPSGTGHLPGLNPL
150 и 134
0,000
0,720
CO6_ALNHLPLEYNSALYSR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
72 и 144
0,019
0,641
VTNC_VDTVDPPYPR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
150 и 135
0,035
0,627
CO6_ALNHLPLEYNSALYSR_vs_VTDB_ELPEHTVK
72 и 147
0,009
0,657
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,043
0,622
CO8A_SLLQPNK_vs_CHL1_VIAVNEVGR
74 и 66
0,007
0,661
VTNC_VDTVDPPYPR_vs_SOM2.CSH_NYGLLYCFR
150 и 138
0,023
0,637
CO8A_SLLQPNK_vs_PSG3_VSAPSGTGHLPGLNPL
74 и 134
0,005
0,671
VTNC_VDTVDPPYPR_vs_SOM2.CSH_SVEGSCGF
150 и 139
0,023
0,639
CO8B_QALEEFQK_vs_CHL1_VIAVNEVGR
76 и 66
0,019
0,642
VTNC_VDTVDPPYPR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
150 и 144
0,043
0,622
CO8B_QALEEFQK_vs_CSH_AHQLAIDTYQEFEETYIPK
76 и 80
0,046
0,620
VTNC_VDTVDPPYPR_vs_VTDB_ELPEHTVK
150 и 147
0,003
0,677
Таблица 28: эффективность классификации по обращениям, недели 17 и 18.
AUROC обращения для недель беременности 17 и 18 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, с BMI-стратификацией (>22≤37).
Обращение
SEQ ID №
p-зн,
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
AFAM_DADPDTFFAK_vs_CHL1_VIAVNEVGR
37 и 66
0,040
0,647
HABP2_FLNWIK_vs_FBLN1_TGYYFDGISR
92 и 86
0,032
0,654
AFAM_DADPDTFFAK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
37 и 144
0,030
0,656
HABP2_FLNWIK_vs_IBP3_FLNVLSPR
92 и 99
0,040
0,647
AFAM_HFQNLGK_vs_CHL1_VIAVNEVGR
38 и 66
0,022
0,664
HABP2_FLNWIK_vs_IGF2_GIVEECCFR
92 и 103
0,018
0,670
ANGT_DPTFIPAPIQAK_vs_CHL1_VIAVNEVGR
42 и 66
0,017
0,671
HABP2_FLNWIK_vs_ITIH4_ILDDLSPR
92 и 112
0,030
0,655
ANGT_DPTFIPAPIQAK_vs_CSH_AHQLAIDTYQEFEETYIPK
42 и 80
0,037
0,650
HABP2_FLNWIK_vs_LYAM1_SYYWIGIR
92 и 120
0,017
0,671
ANGT_DPTFIPAPIQAK_vs_NCAM1_GLGEISAASEFK
42 и 121
0,043
0,645
HABP2_FLNWIK_vs_NCAM1_GLGEISAASEFK
92 и 121
0,014
0,675
ANGT_DPTFIPAPIQAK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
42 и 144
0,033
0,653
HABP2_FLNWIK_vs_PGRP2_AGLLRPDYALLGHR
92 и 126
0,034
0,652
APOH_ATVVYQGER_vs_CHL1_VIAVNEVGR
48 и 66
0,031
0,655
HABP2_FLNWIK_vs_PSG3_VSAPSGTGHLPGLNPL
92 и 134
0,005
0,699
BGH3_LTLLAPLNSVFK_vs_CHL1_VIAVNEVGR
52 и 66
0,016
0,672
HABP2_FLNWIK_vs_SHBG_IALGGLLFPASNLR
92 и 18
0,034
0,652
C1QB_VPGLYYFTYHASSR_vs_CHL1_VIAVNEVGR
55 и 66
0,019
0,669
HABP2_FLNWIK_vs_SPRL1_VLTHSELAPLR
92 и 140
0,046
0,643
C1QB_VPGLYYFTYHASSR_vs_CSH_AHQLAIDTYQEFEETYIPK
55 и 80
0,038
0,648
HABP2_FLNWIK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
92 и 144
0,003
0,709
C1QB_VPGLYYFTYHASSR_vs_CSH_ISLLLIESWLEPVR
55 и 81
0,046
0,643
HABP2_FLNWIK_vs_VTDB_ELPEHTVK
92 и 147
0,005
0,700
C1QB_VPGLYYFTYHASSR_vs_FBLN1_TGYYFDGISR
55 и 86
0,037
0,650
HEMO_NFPSPVDAAFR_vs_CHL1_VIAVNEVGR
93 и 66
0,020
0,667
C1QB_VPGLYYFTYHASSR_vs_IBP2_LIQGAPTIR
55 и 98
0,042
0,646
IBP4_QCHPALDGQR_vs_CHL1_VIAVNEVGR
2 и 66
0,034
0,652
C1QB_VPGLYYFTYHASSR_vs_LYAM1_SYYWIGIR
55 и 120
0,043
0,645
KNG1_DIPTNSPELEETLTHTITK_vs_CHL1_VIAVNEVGR
116 и 66
0,022
0,664
C1QB_VPGLYYFTYHASSR_vs_PGRP2_AGLLRPDYALLGHR
55 и 126
0,030
0,656
PEDF_LQSLFDSPDFSK_vs_CHL1_VIAVNEVGR
124 и 66
0,008
0,691
C1QB_VPGLYYFTYHASSR_vs_SHBG_IALGGLLFPASNLR
55 и 18
0,037
0,650
PEDF_TVQAVLTVPK_vs_CHL1_VIAVNEVGR
125 и 66
0,009
0,688
C1QB_VPGLYYFTYHASSR_vs_SOM2.CSH_NYGLLYCFR
55 и 138
0,029
0,657
PEDF_TVQAVLTVPK_vs_VTDB_ELPEHTVK
125 и 147
0,049
0,641
C1QB_VPGLYYFTYHASSR_vs_SPRL1_VLTHSELAPLR
55 и 140
0,034
0,652
PSG2_IHPSYTNYR_vs_CHL1_VIAVNEVGR
133 и 66
0,021
0,665
CBPN_EALIQFLEQVHQGIK_vs_CHL1_VIAVNEVGR
59 и 66
0,047
0,642
PSG2_IHPSYTNYR_vs_FBLN1_TGYYFDGISR
133 и 86
0,012
0,680
CD14_SWLAELQQWLKPGLK_vs_CHL1_VIAVNEVGR
62 и 66
0,031
0,655
PSG2_IHPSYTNYR_vs_LYAM1_SYYWIGIR
133 и 120
0,025
0,660
CFAB_YGLVTYATYPK_vs_CHL1_VIAVNEVGR
64 и 66
0,006
0,697
PSG2_IHPSYTNYR_vs_NCAM1_GLGEISAASEFK
133 и 121
0,048
0,642
CFAB_YGLVTYATYPK_vs_NCAM1_GLGEISAASEFK
64 и 121
0,017
0,671
PSG2_IHPSYTNYR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
133 и 135
0,012
0,679
CFAB_YGLVTYATYPK_vs_PSG3_VSAPSGTGHLPGLNPL
64 и 134
0,021
0,666
PSG2_IHPSYTNYR_vs_PSG9_LFIPQITR
133 и 136
0,033
0,653
CFAB_YGLVTYATYPK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
64 и 144
0,038
0,648
PSG2_IHPSYTNYR_vs_VTDB_ELPEHTVK
133 и 147
0,043
0,645
CLUS_ASSIIDELFQDR_vs_CHL1_VIAVNEVGR
67 и 66
0,014
0,675
VTNC_GQYCYELDEK_vs_C163A_INPASLDK
149 и 54
0,044
0,644
CLUS_LFDSDPITVTVPVEVSR_vs_CHL1_VIAVNEVGR
68 и 66
0,022
0,664
VTNC_GQYCYELDEK_vs_CHL1_VIAVNEVGR
149 и 66
0,001
0,744
CO5_TLLPVSKPEIR_vs_CHL1_VIAVNEVGR
70 и 66
0,006
0,696
VTNC_GQYCYELDEK_vs_FBLN1_TGYYFDGISR
149 и 86
0,038
0,648
CO5_TLLPVSKPEIR_vs_LYAM1_SYYWIGIR
70 и 120
0,044
0,644
VTNC_GQYCYELDEK_vs_IBP3_FLNVLSPR
149 и 99
0,030
0,655
CO5_TLLPVSKPEIR_vs_PSG3_VSAPSGTGHLPGLNPL
70 и 134
0,038
0,648
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,016
0,672
CO5_VFQFLEK_vs_CHL1_VIAVNEVGR
71 и 66
0,005
0,701
VTNC_GQYCYELDEK_vs_NCAM1_GLGEISAASEFK
149 и 121
0,020
0,667
CO5_VFQFLEK_vs_PSG3_VSAPSGTGHLPGLNPL
71 и 134
0,048
0,642
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,014
0,677
CO5_VFQFLEK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
71 и 144
0,038
0,648
VTNC_GQYCYELDEK_vs_PSG3_VSAPSGTGHLPGLNPL
149 и 134
0,009
0,688
CO6_ALNHLPLEYNSALYSR_vs_CHL1_VIAVNEVGR
72 и 66
0,001
0,741
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,045
0,643
CO6_ALNHLPLEYNSALYSR_vs_FBLN1_TGYYFDGISR
72 и 86
0,045
0,643
VTNC_GQYCYELDEK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
149 и 144
0,004
0,704
CO6_ALNHLPLEYNSALYSR_vs_LYAM1_SYYWIGIR
72 и 120
0,036
0,650
VTNC_GQYCYELDEK_vs_VTDB_ELPEHTVK
149 и 147
0,003
0,712
CO6_ALNHLPLEYNSALYSR_vs_PSG3_VSAPSGTGHLPGLNPL
72 и 134
0,028
0,658
VTNC_VDTVDPPYPR_vs_CHL1_VIAVNEVGR
150 и 66
0,002
0,726
CO6_ALNHLPLEYNSALYSR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
72 и 144
0,007
0,693
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,023
0,663
CO6_ALNHLPLEYNSALYSR_vs_VTDB_ELPEHTVK
72 и 147
0,041
0,647
VTNC_VDTVDPPYPR_vs_PSG3_VSAPSGTGHLPGLNPL
150 и 134
0,023
0,664
F13B_GDTYPAELYITGSILR_vs_CHL1_VIAVNEVGR
84 и 66
0,021
0,665
VTNC_VDTVDPPYPR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
150 и 144
0,028
0,657
HABP2_FLNWIK_vs_CHL1_VIAVNEVGR
92 и 66
0,001
0,729
VTNC_VDTVDPPYPR_vs_VTDB_ELPEHTVK
150 и 147
0,009
0,688
Таблица 29: эффективность классификации по обращениям, недели 17 и 18.
AUROC обращения для недель беременности 17 и 18 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, без BMI-стратификации.
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,021
0,744
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_VTDB_ELPEHTVK
82 и 147
0,008
0,781
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,023
0,741
ENPP2_TYLHTYESEI_vs_ALS_IRPHTFTGLSGLR
83 и 40
0,026
0,735
ANGT_DPTFIPAPIQAK_vs_IGF2_GIVEECCFR
42 и 103
0,017
0,752
ENPP2_TYLHTYESEI_vs_ITIH4_ILDDLSPR
83 и 112
0,028
0,732
CLUS_ASSIIDELFQDR_vs_CHL1_VIAVNEVGR
67 и 66
0,018
0,749
ENPP2_TYLHTYESEI_vs_NCAM1_GLGEISAASEFK
83 и 121
0,021
0,744
CLUS_ASSIIDELFQDR_vs_IBP3_FLNVLSPR
67 и 99
0,043
0,713
ENPP2_TYLHTYESEI_vs_PGRP2_AGLLRPDYALLGHR
83 и 126
0,013
0,762
CLUS_ASSIIDELFQDR_vs_IGF2_GIVEECCFR
67 и 103
0,005
0,795
ENPP2_TYLHTYESEI_vs_PSG1_FQLPGQK
83 и 131
0,021
0,744
CLUS_LFDSDPITVTVPVEVSR_vs_CHL1_VIAVNEVGR
68 и 66
0,046
0,710
ENPP2_TYLHTYESEI_vs_PSG9_LFIPQITR
83 и 136
0,034
0,723
CLUS_LFDSDPITVTVPVEVSR_vs_IGF2_GIVEECCFR
68 и 103
0,012
0,765
ENPP2_TYLHTYESEI_vs_TENX_LSQLSVTDVTTSSLR
83 и 142
0,038
0,719
CO5_TLLPVSKPEIR_vs_IGF2_GIVEECCFR
70 и 103
0,048
0,709
ENPP2_TYLHTYESEI_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
83 и 144
0,031
0,728
CO6_ALNHLPLEYNSALYSR_vs_CHL1_VIAVNEVGR
72 и 66
0,018
0,750
ENPP2_TYLHTYESEI_vs_VTDB_ELPEHTVK
83 и 147
0,010
0,770
CO6_ALNHLPLEYNSALYSR_vs_IBP3_YGQPLPGYTTK
72 и 100
0,047
0,710
HABP2_FLNWIK_vs_CHL1_VIAV NEVGR
92 и 66
0,014
0,758
CO6_ALNHLPLEYNSALYSR_vs_IGF2_GIVEECCFR
72 и 103
0,007
0,787
HABP2_FLNWIK_vs_IBP3_FLNV LSPR
92 и 99
0,040
0,717
CO6_ALNHLPLEYNSALYSR_vs_PSG3_VSAPSGTGHLPGLNPL
72 и 134
0,040
0,717
HABP2_FLNWIK_vs_IBP3_YGQPLPGYTTK
92 и 100
0,043
0,714
CO6_ALNHLPLEYNSALYSR_vs_VTDB_ELPEHTVK
72 и 147
0,034
0,724
HABP2_FLNWIK_vs_IGF2_GIVEE CCFR
92 и 103
0,006
0,790
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_ALS_IRPHTFTGLSGLR
82 и 40
0,012
0,766
HABP2_FLNWIK_vs_ITIH4_ILDD LSPR
92 и 112
0,043
0,714
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_CSH_ISLLLIESWLEPVR
82 и 81
0,037
0,721
HABP2_FLNWIK_vs_PSG3_VSAPSGTGHLPGLNPL
92 и 134
0,028
0,732
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_FBLN1_TGYYFDGISR
82 и 86
0,026
0,735
HABP2_FLNWIK_vs_SOM2.CSH_SVEGSCGF
92 и 139
0,047
0,710
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_IBP3_FLNVLSPR
82 и 99
0,025
0,737
KNG1_QVVAGLNFR_vs_IGF2_GIVEECCFR
117 и 103
0,020
0,746
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_IBP3_YGQPLPGYTTK
82 и 100
0,029
0,731
PSG2_IHPSYTNYR_vs_C163A_INPASLDK
133 и 54
0,044
0,712
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_ITIH4_ILDDLSPR
82 и 112
0,010
0,773
PSG2_IHPSYTNYR_vs_CHL1_VIAVNEVGR
133 и 66
0,026
0,734
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_NCAM1_GLGEISAASEFK
82 и 121
0,007
0,785
PSG2_IHPSYTNYR_vs_CRIS3_AVSPPAR
133 и 78
0,025
0,736
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PGRP2_AGLLRPDYALLGHR
82 и 126
0,012
0,767
PSG2_IHPSYTNYR_vs_CRIS3_YEDLYSNCK
133 и 79
0,017
0,751
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PSG1_FQLPGQK
82 и 131
0,011
0,769
PSG2_IHPSYTNYR_vs_CSH_AHQLAIDTYQEFEETYIPK
133 и 80
0,034
0,723
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
82 и 135
0,043
0,713
PSG2_IHPSYTNYR_vs_IGF2_GIVEECCFR
133 и 103
0,018
0,750
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PSG9_LFIPQITR
82 и 136
0,018
0,749
PSG2_IHPSYTNYR_vs_LYAM1_SYYWIGIR
133 и 120
0,031
0,728
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_SPRL1_VLTHSELAPLR
82 и 140
0,035
0,722
PSG2_IHPSYTNYR_vs_PSG3_VSAPSGTGHLPGLNPL
133 и 134
0,032
0,727
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_TENX_LNWEAPPGAFDSFLLR
82 и 141
0,046
0,710
PSG2_IHPSYTNYR_vs_SOM2.CSH_NYGLLYCFR
133 и 138
0,044
0,712
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_TENX_LSQLSVTDVTTSSLR
82 и 142
0,024
0,739
PSG2_IHPSYTNYR_vs_SOM2.CSH_SVEGSCGF
133 и 139
0,028
0,731
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
82 и 144
0,016
0,754
VTNC_GQYCYELDEK_vs_IGF2_GIVEECCFR
149 и 103
0,032
0,726
Таблица 30: эффективность классификации по обращениям, недели 17 и 18.
AUROC обращения для недель беременности 17 и 18 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, с BMI-стратификацией (>22≤37).
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,042
0,749
ENPP2_TYLHTYESEI_vs_TENX_LSQLSVTDVTTSSLR
83 и 142
0,033
0,762
ANGT_DPTFIPAPIQAK_vs_IBP3_FLNVLSPR
42 и 99
0,033
0,762
ENPP2_TYLHTYESEI_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
83 и 144
0,021
0,784
ANGT_DPTFIPAPIQAK_vs_IBP3_YGQPLPGYTTK
42 и 100
0,041
0,751
ENPP2_TYLHTYESEI_vs_VTDB_ELPEHTVK
83 и 147
0,011
0,811
ANGT_DPTFIPAPIQAK_vs_IGF2_GIVEECCFR
42 и 103
0,017
0,793
F13B_GDTYPAELYITGSILR_vs_IGF2_GIVEECCFR
84 и 103
0,034
0,760
B2MG_VNHVTLSQPK_vs_IGF2_GIVEECCFR
51 и 103
0,042
0,749
HABP2_FLNWIK_vs_CHL1_VIAVNEVGR
92 и 66
0,037
0,756
CD14_LTVGAAQVPAQLLVGALR_vs_IGF2_GIVEECCFR
61 и 103
0,042
0,749
HABP2_FLNWIK_vs_IBP3_FLNVLSPR
92 и 99
0,016
0,797
CLUS_ASSIIDELFQDR_vs_IGF2_GIVEECCFR
67 и 103
0,020
0,786
HABP2_FLNWIK_vs_IBP3_YGQPLPGYTTK
92 и 100
0,032
0,764
CLUS_LFDSDPITVTVPVEVSR_vs_IGF2_GIVEECCFR
68 и 103
0,047
0,744
HABP2_FLNWIK_vs_IGF2_GIVEECCFR
92 и 103
0,005
0,842
CO5_TLLPVSKPEIR_vs_IGF2_GIVEECCFR
70 и 103
0,039
0,753
HABP2_FLNWIK_vs_LYAM1_SYYWIGIR
92 и 120
0,028
0,769
CO6_ALNHLPLEYNSALYSR_vs_CHL1_VIAVNEVGR
72 и 66
0,026
0,773
HABP2_FLNWIK_vs_SOM2.CSH_SVEGSCGF
92 и 139
0,045
0,746
CO6_ALNHLPLEYNSALYSR_vs_IBP3_FLNVLSPR
72 и 99
0,016
0,795
ITIH4_QLGLPGPPDVPDHAAYHPF_vs_CRIS3_YEDLYSNCK
114 и 79
0,046
0,745
CO6_ALNHLPLEYNSALYSR_vs_IBP3_YGQPLPGYTTK
72 и 100
0,025
0,775
KNG1_DIPTNSPELEETLTHTITK_vs_IGF2_GIVEECCFR
116 и 103
0,033
0,762
CO6_ALNHLPLEYNSALYSR_vs_IGF2_GIVEECCFR
72 и 103
0,005
0,842
KNG1_QVVAGLNFR_vs_IBP3_FLNVLSPR
117 и 99
0,018
0,791
CO6_ALNHLPLEYNSALYSR_vs_LYAM1_SYYWIGIR
72 и 120
0,025
0,775
KNG1_QVVAGLNFR_vs_IBP3_YGQPLPGYTTK
117 и 100
0,027
0,771
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_ALS_IRPHTFTGLSGLR
82 и 40
0,041
0,751
KNG1_QVVAGLNFR_vs_IGF2_GIVEECCFR
117 и 103
0,006
0,839
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_CSH_ISLLLIESWLEPVR
82 и 81
0,029
0,767
PSG2_IHPSYTNYR_vs_C163A_INPASLDK
133 и 54
0,030
0,766
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_ITIH4_ILDDLSPR
82 и 112
0,019
0,788
PSG2_IHPSYTNYR_vs_CHL1_VIAVNEVGR
133 и 66
0,023
0,778
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_NCAM1_GLGEISAASEFK
82 и 121
0,022
0,782
PSG2_IHPSYTNYR_vs_CRIS3_AVSPPAR
133 и 78
0,008
0,826
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PGRP2_AGLLRPDYALLGHR
82 и 126
0,026
0,773
PSG2_IHPSYTNYR_vs_CRIS3_YEDLYSNCK
133 и 79
0,010
0,817
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PSG1_FQLPGQK
82 и 131
0,039
0,753
PSG2_IHPSYTNYR_vs_CSH_AHQLAIDTYQEFEETYIPK
133 и 80
0,037
0,756
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_SHBG_IALGGLLFPASNLR
82 и 18
0,044
0,747
PSG2_IHPSYTNYR_vs_FBLN1_TGYYFDGISR
133 и 86
0,014
0,800
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_SOM2.CSH_NYGLLYCFR
82 и 138
0,049
0,742
PSG2_IHPSYTNYR_vs_IBP3_FLNVLSPR
133 и 99
0,015
0,799
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_TENX_LSQLSVTDVTTSSLR
82 и 142
0,029
0,767
PSG2_IHPSYTNYR_vs_IBP3_YGQPLPGYTTK
133 и 100
0,022
0,780
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
82 и 144
0,015
0,799
PSG2_IHPSYTNYR_vs_IGF2_GIVEECCFR
133 и 103
0,005
0,842
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_VTDB_ELPEHTVK
82 и 147
0,014
0,802
PSG2_IHPSYTNYR_vs_LYAM1_SYYWIGIR
133 и 120
0,008
0,828
ENPP2_TYLHTYESEI_vs_CSH_ISLLLIESWLEPVR
83 и 81
0,047
0,744
PSG2_IHPSYTNYR_vs_PSG3_VSAPSGTGHLPGLNPL
133 и 134
0,028
0,769
ENPP2_TYLHTYESEI_vs_ITIH4_ILDDLSPR
83 и 112
0,038
0,755
PSG2_IHPSYTNYR_vs_SOM2.CSH_SVEGSCGF
133 и 139
0,040
0,752
ENPP2_TYLHTYESEI_vs_NCAM1_GLGEISAASEFK
83 и 121
0,034
0,760
THBG_AVLHIGEK_vs_IGF2_GIVEECCFR
143 и 103
0,042
0,749
ENPP2_TYLHTYESEI_vs_PGRP2_AGLLRPDYALLGHR
83 и 126
0,033
0,762
VTNC_GQYCYELDEK_vs_IBP3_FLNVLSPR
149 и 99
0,042
0,749
ENPP2_TYLHTYESEI_vs_SHBG_IALGGLLFPASNLR
83 и 18
0,044
0,747
VTNC_GQYCYELDEK_vs_IGF2_GIVEECCFR
149 и 103
0,015
0,799
Таблица 31: эффективность классификации по обращениям, недели 18 и 19.
AUROC обращения для недель беременности 18 и 19 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, без BMI-стратификации.
Обращение
SEQ ID №
p-зн.
ROC_AUC
Обращение
SEQ ID №
p-зн,
ROC_AUC
A2GL_DLLLPQPDLR_vs_CSH_AHQLAIDTYQEFEETYIPK
34 и 80
0,030
0,603
CO8A_SLLQPNK_vs_PSG3_VSAPSGTGHLPGLNPL
74 и 134
0,007
0,627
A2GL_DLLLPQPDLR_vs_FBLN1_TGYYFDGISR
34 и 86
0,016
0,613
CO8A_SLLQPNK_vs_TENX_LNWEAPPGAFDSFLLR
74 и 141
0,021
0,609
A2GL_DLLLPQPDLR_vs_PSG3_VSAPSGTGHLPGLNPL
34 и 134
0,003
0,642
CO8A_SLLQPNK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
74 и 144
0,001
0,652
A2GL_DLLLPQPDLR_vs_SHBG_IALGGLLFPASNLR
34 и 18
0,015
0,615
CO8B_QALEEFQK_vs_CSH_AHQLAIDTYQEFEETYIPK
76 и 80
0,016
0,614
A2GL_DLLLPQPDLR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
34 и 144
0,024
0,607
CO8B_QALEEFQK_vs_FBLN1_TGYYFDGISR
76 и 86
0,034
0,600
AFAM_DADPDTFFAK_vs_FBLN1_TGYYFDGISR
37 и 86
0,034
0,600
CO8B_QALEEFQK_vs_PSG3_VSAPSGTGHLPGLNPL
76 и 134
0,007
0,627
AFAM_DADPDTFFAK_vs_PSG3_VSAPSGTGHLPGLNPL
37 и 134
0,015
0,615
CO8B_QALEEFQK_vs_TENX_LNWEAPPGAFDSFLLR
76 и 141
0,019
0,611
AFAM_DADPDTFFAK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
37 и 144
0,009
0,623
CO8B_QALEEFQK_vs_TENX_LSQLSVTDVTTSSLR
76 и 142
0,024
0,607
AFAM_HFQNLGK_vs_PSG3_VSAPSGTGHLPGLNPL
38 и 134
0,025
0,606
CO8B_QALEEFQK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
76 и 144
0,001
0,654
AFAM_HFQNLGK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
38 и 144
0,021
0,609
F13B_GDTYPAELYITGSILR_vs_FBLN1_TGYYFDGISR
84 и 86
0,011
0,620
ANGT_DPTFIPAPIQAK_vs_CSH_AHQLAIDTYQEFEETYIPK
42 и 80
0,014
0,616
F13B_GDTYPAELYITGSILR_vs_PSG3_VSAPSGTGHLPGLNPL
84 и 134
0,005
0,631
ANGT_DPTFIPAPIQAK_vs_FBLN1_TGYYFDGISR
42 и 86
0,008
0,625
F13B_GDTYPAELYITGSILR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
84 и 144
0,023
0,607
ANGT_DPTFIPAPIQAK_vs_PSG3_VSAPSGTGHLPGLNPL
42 и 134
0,005
0,633
FBLN3_IPSNPSHR_vs_SHBG_IALGGLLFPASNLR
87 и 18
0,031
0,602
ANGT_DPTFIPAPIQAK_vs_SHBG_IALGGLLFPASNLR
42 и 18
0,018
0,612
FETUA_FSVVYAK_vs_PSG3_VSAPSGTGHLPGLNPL
88 и 134
0,025
0,606
ANGT_DPTFIPAPIQAK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
42 и 144
0,010
0,621
FETUA_FSVVYAK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
88 и 144
0,033
0,601
APOC3_GWVTDGFSSLK_vs_ALS_IRPHTFTGLSGLR
47 и 40
0,018
0,612
FETUA_HTLNQIDEVK_vs_PSG3_VSAPSGTGHLPGLNPL
89 и 134
0,024
0,607
APOC3_GWVTDGFSSLK_vs_C163A_INPASLDK
47 и 54
0,002
0,648
FETUA_HTLNQIDEVK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
89 и 144
0,033
0,601
APOC3_GWVTDGFSSLK_vs_CHL1_VIAVNEVGR
47 и 66
0,033
0,601
HABP2_FLNWIK_vs_FBLN1_TGYYFDGISR
92 и 86
0,013
0,617
APOC3_GWVTDGFSSLK_vs_CRIS3_AVSPPAR
47 и 78
0,022
0,608
HABP2_FLNWIK_vs_PSG3_VSAPSGTGHLPGLNPL
92 и 134
0,004
0,635
APOC3_GWVTDGFSSLK_vs_CRIS3_YEDLYSNCK
47 и 79
0,014
0,616
HABP2_FLNWIK_vs_TENX_LNWEAPPGAFDSFLLR
92 и 141
0,024
0,607
APOC3_GWVTDGFSSLK_vs_CSH_AHQLAIDTYQEFEETYIPK
47 и 80
0,007
0,627
HABP2_FLNWIK_vs_TENX_LSQLSVTDVTTSSLR
92 и 142
0,029
0,603
APOC3_GWVTDGFSSLK_vs_CSH_ISLLLIESWLEPVR
47 и 81
0,022
0,608
HABP2_FLNWIK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
92 и 144
0,010
0,622
APOC3_GWVTDGFSSLK_vs_FBLN1_TGYYFDGISR
47 и 86
0,012
0,619
HEMO_NFPSPVDAAFR_vs_FBLN1_TGYYFDGISR
93 и 86
0,024
0,607
APOC3_GWVTDGFSSLK_vs_IBP3_FLNVLSPR
47 и 99
0,017
0,613
HEMO_NFPSPVDAAFR_vs_PSG3_VSAPSGTGHLPGLNPL
93 и 134
0,008
0,624
APOC3_GWVTDGFSSLK_vs_IBP3_YGQPLPGYTTK
47 и 100
0,026
0,605
HEMO_NFPSPVDAAFR_vs_SHBG_IALGGLLFPASNLR
93 и 18
0,030
0,602
APOC3_GWVTDGFSSLK_vs_IGF2_GIVEECCFR
47 и 103
0,016
0,614
HEMO_NFPSPVDAAFR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
93 и 144
0,011
0,620
APOC3_GWVTDGFSSLK_vs_ITIH4_ILDDLSPR
47 и 112
0,032
0,601
IBP4_QCHPALDGQR_vs_CSH_AHQLAIDTYQEFEETYIPK
2 и 80
0,025
0,606
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,009
0,623
IBP4_QCHPALDGQR_vs_FBLN1_TGYYFDGISR
2 и 86
0,004
0,636
APOC3_GWVTDGFSSLK_vs_NCAM1_GLGEISAASEFK
47 и 121
0,028
0,604
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,033
0,601
APOC3_GWVTDGFSSLK_vs_PGRP2_AGLLRPDYALLGHR
47 и 126
0,026
0,605
IBP4_QCHPALDGQR_vs_PSG3_VSAPSGTGHLPGLNPL
2 и 134
0,000
0,668
APOC3_GWVTDGFSSLK_vs_PRG2_WNFAYWAAHQPWSR
47 и 129
0,024
0,607
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,011
0,621
APOC3_GWVTDGFSSLK_vs_PSG3_VSAPSGTGHLPGLNPL
47 и 134
0,002
0,644
IBP4_QCHPALDGQR_vs_TENX_LNWEAPPGAFDSFLLR
2 и 141
0,028
0,603
APOC3_GWVTDGFSSLK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
47 и 135
0,034
0,600
IBP4_QCHPALDGQR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
2 и 144
0,006
0,629
APOC3_GWVTDGFSSLK_vs_SHBG_IALGGLLFPASNLR
47 и 18
0,018
0,611
IBP6_GAQTLYVPNCDHR_vs_FBLN1_TGYYFDGISR
101 и 86
0,026
0,605
APOC3_GWVTDGFSSLK_vs_SOM2.CSH_NYGLLYCFR
47 и 138
0,022
0,608
IBP6_GAQTLYVPNCDHR_vs_PSG3_VSAPSGTGHLPGLNPL
101 и 134
0,014
0,617
APOC3_GWVTDGFSSLK_vs_SOM2.CSH_SVEGSCGF
47 и 139
0,025
0,607
IBP6_HLDSVLQQLQTEVYR_vs_FBLN1_TGYYFDGISR
102 и 86
0,023
0,608
APOC3_GWVTDGFSSLK_vs_TENX_LNWEAPPGAFDSFLLR
47 и 141
0,018
0,612
IBP6_HLDSVLQQLQTEVYR_vs_PSG3_VSAPSGTGHLPGLNPL
102 и 134
0,014
0,617
APOC3_GWVTDGFSSLK_vs_TENX_LSQLSVTDVTTSSLR
47 и 142
0,017
0,612
INHBC_LDFHFSSDR_vs_C163A_INPASLDK
107 и 54
0,013
0,617
APOC3_GWVTDGFSSLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
47 и 144
0,005
0,632
INHBC_LDFHFSSDR_vs_FBLN1_TGYYFDGISR
107 и 86
0,016
0,614
APOC3_GWVTDGFSSLK_vs_VTDB_ELPEHTVK
47 и 147
0,025
0,606
INHBC_LDFHFSSDR_vs_PSG3_VSAPSGTGHLPGLNPL
107 и 134
0,005
0,634
APOH_ATVVYQGER_vs_ALS_IRPHTFTGLSGLR
48 и 40
0,034
0,600
INHBC_LDFHFSSDR_vs_TENX_LNWEAPPGAFDSFLLR
107 и 141
0,012
0,618
APOH_ATVVYQGER_vs_C163A_INPASLDK
48 и 54
0,023
0,607
INHBC_LDFHFSSDR_vs_TENX_LSQLSVTDVTTSSLR
107 и 142
0,020
0,610
APOH_ATVVYQGER_vs_CSH_AHQLAIDTYQEFEETYIPK
48 и 80
0,011
0,620
INHBC_LDFHFSSDR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
107 и 144
0,011
0,620
APOH_ATVVYQGER_vs_FBLN1_TGYYFDGISR
48 и 86
0,002
0,645
ITIH3_ALDLSLK_vs_PSG3_VSAPSGTGHLPGLNPL
111 и 134
0,033
0,601
APOH_ATVVYQGER_vs_IGF2_GIVEECCFR
48 и 103
0,024
0,607
ITIH3_ALDLSLK_vs_SHBG_IALGGLLFPASNLR
111 и 18
0,024
0,606
APOH_ATVVYQGER_vs_LYAM1_SYYWIGIR
48 и 120
0,017
0,613
ITIH4_NPLVWVHASPEHVVVTR_vs_FBLN1_TGYYFDGISR
113 и 86
0,018
0,612
APOH_ATVVYQGER_vs_PGRP2_AGLLRPDYALLGHR
48 и 126
0,025
0,606
ITIH4_NPLVWVHASPEHVVVTR_vs_PSG3_VSAPSGTGHLPGLNPL
113 и 134
0,015
0,615
APOH_ATVVYQGER_vs_PSG3_VSAPSGTGHLPGLNPL
48 и 134
0,001
0,664
ITIH4_QLGLPGPPDVPDHAAYHPF_vs_PSG3_VSAPSGTGHLPGLNPL
114 и 134
0,029
0,604
APOH_ATVVYQGER_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
48 и 135
0,030
0,603
KNG1_DIPTNSPELEETLTHTITK_vs_CSH_AHQLAIDTYQEFEETYIPK
116 и 80
0,023
0,607
APOH_ATVVYQGER_vs_SHBG_IALGGLLFPASNLR
48 и 18
0,008
0,625
KNG1_DIPTNSPELEETLTHTITK_vs_FBLN1_TGYYFDGISR
116 и 86
0,018
0,612
APOH_ATVVYQGER_vs_TENX_LNWEAPPGAFDSFLLR
48 и 141
0,016
0,614
KNG1_DIPTNSPELEETLTHTITK_vs_PSG3_VSAPSGTGHLPGLNPL
116 и 134
0,005
0,634
APOH_ATVVYQGER_vs_TENX_LSQLSVTDVTTSSLR
48 и 142
0,013
0,617
KNG1_DIPTNSPELEETLTHTITK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
116 и 144
0,010
0,621
APOH_ATVVYQGER_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
48 и 144
0,002
0,645
KNG1_QVVAGLNFR_vs_FBLN1_TGYYFDGISR
117 и 86
0,022
0,608
APOH_ATVVYQGER_vs_VTDB_ELPEHTVK
48 и 147
0,030
0,602
KNG1_QVVAGLNFR_vs_PSG3_VSAPSGTGHLPGLNPL
117 и 134
0,014
0,616
B2MG_VNHVTLSQPK_vs_PSG3_VSAPSGTGHLPGLNPL
51 и 134
0,019
0,611
KNG1_QVVAGLNFR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
117 и 144
0,032
0,601
C1QB_VPGLYYFTYHASSR_vs_CSH_AHQLAIDTYQEFEETYIPK
55 и 80
0,006
0,630
LBP_ITGFLKPGK_vs_C163A_INPASLDK
118 и 54
0,007
0,627
C1QB_VPGLYYFTYHASSR_vs_CSH_ISLLLIESWLEPVR
55 и 81
0,020
0,610
LBP_ITGFLKPGK_vs_PSG3_VSAPSGTGHLPGLNPL
118 и 134
0,002
0,646
C1QB_VPGLYYFTYHASSR_vs_FBLN1_TGYYFDGISR
55 и 86
0,006
0,631
LBP_ITGFLKPGK_vs_SHBG_IALGGLLFPASNLR
118 и 18
0,019
0,610
C1QB_VPGLYYFTYHASSR_vs_LYAM1_SYYWIGIR
55 и 120
0,030
0,603
LBP_ITGFLKPGK_vs_SOM2.CSH_SVEGSCGF
118 и 139
0,032
0,602
C1QB_VPGLYYFTYHASSR_vs_PGRP2_AGLLRPDYALLGHR
55 и 126
0,024
0,606
LBP_ITGFLKPGK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
118 и 144
0,022
0,608
C1QB_VPGLYYFTYHASSR_vs_PSG3_VSAPSGTGHLPGLNPL
55 и 134
0,006
0,630
LBP_ITLPDFTGDLR_vs_C163A_INPASLDK
119 и 54
0,004
0,635
C1QB_VPGLYYFTYHASSR_vs_SHBG_IALGGLLFPASNLR
55 и 18
0,006
0,629
LBP_ITLPDFTGDLR_vs_CHL1_VIAVNEVGR
119 и 66
0,027
0,605
C1QB_VPGLYYFTYHASSR_vs_SOM2.CSH_NYGLLYCFR
55 и 138
0,016
0,613
LBP_ITLPDFTGDLR_vs_CSH_AHQLAIDTYQEFEETYIPK
119 и 80
0,014
0,616
C1QB_VPGLYYFTYHASSR_vs_TENX_LNWEAPPGAFDSFLLR
55 и 141
0,028
0,604
LBP_ITLPDFTGDLR_vs_FBLN1_TGYYFDGISR
119 и 86
0,017
0,612
C1QB_VPGLYYFTYHASSR_vs_TENX_LSQLSVTDVTTSSLR
55 и 142
0,020
0,610
LBP_ITLPDFTGDLR_vs_LYAM1_SYYWIGIR
119 и 120
0,031
0,602
C1QB_VPGLYYFTYHASSR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
55 и 144
0,013
0,618
LBP_ITLPDFTGDLR_vs_PSG3_VSAPSGTGHLPGLNPL
119 и 134
0,001
0,652
CBPN_EALIQFLEQVHQGIK_vs_FBLN1_TGYYFDGISR
59 и 86
0,030
0,603
LBP_ITLPDFTGDLR_vs_SHBG_IALGGLLFPASNLR
119 и 18
0,013
0,618
CD14_LTVGAAQVPAQLLVGALR_vs_CSH_AHQLAIDTYQEFEETYIPK
61 и 80
0,017
0,613
LBP_ITLPDFTGDLR_vs_SOM2.CSH_SVEGSCGF
119 и 139
0,018
0,612
CD14_LTVGAAQVPAQLLVGALR_vs_FBLN1_TGYYFDGISR
61 и 86
0,010
0,622
LBP_ITLPDFTGDLR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
119 и 144
0,015
0,615
CD14_LTVGAAQVPAQLLVGALR_vs_LYAM1_SYYWIGIR
61 и 120
0,025
0,606
LBP_ITLPDFTGDLR_vs_VTDB_ELPEHTVK
119 и 147
0,034
0,600
CD14_LTVGAAQVPAQLLVGALR_vs_PSG3_VSAPSGTGHLPGLNPL
61 и 134
0,003
0,641
PEDF_LQSLFDSPDFSK_vs_C163A_INPASLDK
124 и 54
0,033
0,601
CD14_LTVGAAQVPAQLLVGALR_vs_SHBG_IALGGLLFPASNLR
61 и 18
0,025
0,606
PEDF_LQSLFDSPDFSK_vs_FBLN1_TGYYFDGISR
124 и 86
0,022
0,608
CD14_LTVGAAQVPAQLLVGALR_vs_TENX_LNWEAPPGAFDSFLLR
61 и 141
0,021
0,609
PEDF_LQSLFDSPDFSK_vs_PSG3_VSAPSGTGHLPGLNPL
124 и 134
0,005
0,634
CD14_LTVGAAQVPAQLLVGALR_vs_TENX_LSQLSVTDVTTSSLR
61 и 142
0,019
0,611
PEDF_LQSLFDSPDFSK_vs_TENX_LNWEAPPGAFDSFLLR
124 и 141
0,028
0,604
CD14_LTVGAAQVPAQLLVGALR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
61 и 144
0,004
0,637
PEDF_LQSLFDSPDFSK_vs_TENX_LSQLSVTDVTTSSLR
124 и 142
0,018
0,611
CD14_SWLAELQQWLKPGLK_vs_CSH_AHQLAIDTYQEFEETYIPK
62 и 80
0,024
0,607
PEDF_LQSLFDSPDFSK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
124 и 144
0,014
0,616
CD14_SWLAELQQWLKPGLK_vs_FBLN1_TGYYFDGISR
62 и 86
0,010
0,621
PEDF_TVQAVLTVPK_vs_C163A_INPASLDK
125 и 54
0,021
0,609
CD14_SWLAELQQWLKPGLK_vs_PSG3_VSAPSGTGHLPGLNPL
62 и 134
0,006
0,631
PEDF_TVQAVLTVPK_vs_CSH_AHQLAIDTYQEFEETYIPK
125 и 80
0,027
0,605
CD14_SWLAELQQWLKPGLK_vs_SHBG_IALGGLLFPASNLR
62 и 18
0,026
0,605
PEDF_TVQAVLTVPK_vs_FBLN1_TGYYFDGISR
125 и 86
0,010
0,622
CD14_SWLAELQQWLKPGLK_vs_TENX_LNWEAPPGAFDSFLLR
62 и 141
0,027
0,604
PEDF_TVQAVLTVPK_vs_PSG3_VSAPSGTGHLPGLNPL
125 и 134
0,003
0,643
CD14_SWLAELQQWLKPGLK_vs_TENX_LSQLSVTDVTTSSLR
62 и 142
0,025
0,606
PEDF_TVQAVLTVPK_vs_TENX_LNWEAPPGAFDSFLLR
125 и 141
0,019
0,611
CD14_SWLAELQQWLKPGLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
62 и 144
0,006
0,631
PEDF_TVQAVLTVPK_vs_TENX_LSQLSVTDVTTSSLR
125 и 142
0,018
0,612
CFAB_YGLVTYATYPK_vs_C163A_INPASLDK
64 и 54
0,015
0,615
PEDF_TVQAVLTVPK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
125 и 144
0,007
0,628
CFAB_YGLVTYATYPK_vs_CSH_AHQLAIDTYQEFEETYIPK
64 и 80
0,034
0,600
PTGDS_GPGEDFR_vs_FBLN1_TGYYFDGISR
137 и 86
0,023
0,607
CFAB_YGLVTYATYPK_vs_FBLN1_TGYYFDGISR
64 и 86
0,032
0,601
PTGDS_GPGEDFR_vs_PSG3_VSAPSGTGHLPGLNPL
137 и 134
0,013
0,618
CFAB_YGLVTYATYPK_vs_PSG3_VSAPSGTGHLPGLNPL
64 и 134
0,006
0,631
VTNC_GQYCYELDEK_vs_C163A_INPASLDK
149 и 54
0,014
0,616
CFAB_YGLVTYATYPK_vs_SHBG_IALGGLLFPASNLR
64 и 18
0,028
0,604
VTNC_GQYCYELDEK_vs_CSH_AHQLAIDTYQEFEETYIPK
149 и 80
0,019
0,611
CFAB_YGLVTYATYPK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
64 и 144
0,003
0,642
VTNC_GQYCYELDEK_vs_FBLN1_TGYYFDGISR
149 и 86
0,004
0,638
CLUS_ASSIIDELFQDR_vs_FBLN1_TGYYFDGISR
67 и 86
0,021
0,609
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,007
0,627
CLUS_ASSIIDELFQDR_vs_PSG3_VSAPSGTGHLPGLNPL
67 и 134
0,013
0,618
VTNC_GQYCYELDEK_vs_NCAM1_GLGEISAASEFK
149 и 121
0,031
0,602
CLUS_LFDSDPITVTVPVEVSR_vs_FBLN1_TGYYFDGISR
68 и 86
0,026
0,605
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,012
0,619
CLUS_LFDSDPITVTVPVEVSR_vs_PSG3_VSAPSGTGHLPGLNPL
68 и 134
0,018
0,611
VTNC_GQYCYELDEK_vs_PSG3_VSAPSGTGHLPGLNPL
149 и 134
0,001
0,657
CO5_TLLPVSKPEIR_vs_FBLN1_TGYYFDGISR
70 и 86
0,020
0,610
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,005
0,632
CO5_TLLPVSKPEIR_vs_PSG3_VSAPSGTGHLPGLNPL
70 и 134
0,011
0,620
VTNC_GQYCYELDEK_vs_SOM2.CSH_SVEGSCGF
149 и 139
0,034
0,601
CO5_TLLPVSKPEIR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
70 и 144
0,012
0,618
VTNC_GQYCYELDEK_vs_TENX_LNWEAPPGAFDSFLLR
149 и 141
0,032
0,601
CO5_VFQFLEK_vs_FBLN1_TGYYFDGISR
71 и 86
0,020
0,610
VTNC_GQYCYELDEK_vs_TENX_LSQLSVTDVTTSSLR
149 и 142
0,026
0,605
CO5_VFQFLEK_vs_PSG3_VSAPSGTGHLPGLNPL
71 и 134
0,024
0,607
VTNC_GQYCYELDEK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
149 и 144
0,001
0,655
CO5_VFQFLEK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
71 и 144
0,011
0,620
VTNC_GQYCYELDEK_vs_VTDB_ELPEHTVK
149 и 147
0,028
0,604
CO6_ALNHLPLEYNSALYSR_vs_FBLN1_TGYYFDGISR
72 и 86
0,024
0,607
VTNC_VDTVDPPYPR_vs_FBLN1_TGYYFDGISR
150 и 86
0,014
0,616
CO6_ALNHLPLEYNSALYSR_vs_PSG3_VSAPSGTGHLPGLNPL
72 и 134
0,020
0,610
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,012
0,618
CO6_ALNHLPLEYNSALYSR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
72 и 144
0,006
0,630
VTNC_VDTVDPPYPR_vs_PSG3_VSAPSGTGHLPGLNPL
150 и 134
0,001
0,650
CO8A_SLLQPNK_vs_CSH_AHQLAIDTYQEFEETYIPK
74 и 80
0,020
0,610
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,015
0,615
CO8A_SLLQPNK_vs_FBLN1_TGYYFDGISR
74 и 86
0,021
0,609
VTNC_VDTVDPPYPR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
150 и 144
0,004
0,637
Таблица 32: эффективность классификации по обращениям, недели 18 и 19.
AUROC обращения для недель беременности 18 и 19 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, с BMI-стратификацией (>22≤37).
Обращение
SEQ ID №
p-зн.
ROC_AUC
Обращение
SEQ ID №
p-зн,
ROC_AUC
A2GL_DLLLPQPDLR_vs_C163A_INPASLDK
34 и 54
0,021
0,634
CO6_ALNHLPLEYNSALYSR_vs_TENX_LSQLSVTDVTTSSLR
72 и 142
0,016
0,640
A2GL_DLLLPQPDLR_vs_CRIS3_AVSPPAR
34 и 78
0,049
0,614
CO6_ALNHLPLEYNSALYSR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
72 и 144
0,014
0,642
A2GL_DLLLPQPDLR_vs_CRIS3_YEDLYSNCK
34 и 79
0,041
0,619
CO8A_SLLQPNK_vs_C163A_INPASLDK
74 и 54
0,036
0,622
A2GL_DLLLPQPDLR_vs_CSH_AHQLAIDTYQEFEETYIPK
34 и 80
0,042
0,618
CO8A_SLLQPNK_vs_LYAM1_SYYWIGIR
74 и 120
0,035
0,623
A2GL_DLLLPQPDLR_vs_FBLN1_TGYYFDGISR
34 и 86
0,034
0,623
CO8A_SLLQPNK_vs_PSG3_VSAPSGTGHLPGLNPL
74 и 134
0,030
0,626
A2GL_DLLLPQPDLR_vs_LYAM1_SYYWIGIR
34 и 120
0,011
0,648
CO8A_SLLQPNK_vs_TENX_LNWEAPPGAFDSFLLR
74 и 141
0,007
0,656
A2GL_DLLLPQPDLR_vs_PGRP2_AGLLRPDYALLGHR
34 и 126
0,020
0,636
CO8A_SLLQPNK_vs_TENX_LSQLSVTDVTTSSLR
74 и 142
0,009
0,651
A2GL_DLLLPQPDLR_vs_PSG3_VSAPSGTGHLPGLNPL
34 и 134
0,010
0,651
CO8A_SLLQPNK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
74 и 144
0,005
0,664
A2GL_DLLLPQPDLR_vs_SHBG_IALGGLLFPASNLR
34 и 18
0,018
0,637
CO8B_QALEEFQK_vs_C163A_INPASLDK
76 и 54
0,049
0,614
A2GL_DLLLPQPDLR_vs_SPRL1_VLTHSELAPLR
34 и 140
0,019
0,637
CO8B_QALEEFQK_vs_PSG3_VSAPSGTGHLPGLNPL
76 и 134
0,037
0,621
A2GL_DLLLPQPDLR_vs_TENX_LNWEAPPGAFDSFLLR
34 и 141
0,021
0,634
CO8B_QALEEFQK_vs_SPRL1_VLTHSELAPLR
76 и 140
0,046
0,616
A2GL_DLLLPQPDLR_vs_TENX_LSQLSVTDVTTSSLR
34 и 142
0,018
0,637
CO8B_QALEEFQK_vs_TENX_LNWEAPPGAFDSFLLR
76 и 141
0,005
0,663
A2GL_DLLLPQPDLR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
34 и 144
0,010
0,651
CO8B_QALEEFQK_vs_TENX_LSQLSVTDVTTSSLR
76 и 142
0,006
0,659
AFAM_DADPDTFFAK_vs_C163A_INPASLDK
37 и 54
0,048
0,615
CO8B_QALEEFQK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
76 и 144
0,005
0,664
AFAM_DADPDTFFAK_vs_PSG3_VSAPSGTGHLPGLNPL
37 и 134
0,021
0,634
F13B_GDTYPAELYITGSILR_vs_CHL1_VIAVNEVGR
84 и 66
0,031
0,625
AFAM_DADPDTFFAK_vs_TENX_LNWEAPPGAFDSFLLR
37 и 141
0,030
0,626
F13B_GDTYPAELYITGSILR_vs_CSH_AHQLAIDTYQEFEETYIPK
84 и 80
0,035
0,623
AFAM_DADPDTFFAK_vs_TENX_LSQLSVTDVTTSSLR
37 и 142
0,033
0,624
F13B_GDTYPAELYITGSILR_vs_FBLN1_TGYYFDGISR
84 и 86
0,036
0,622
AFAM_DADPDTFFAK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
37 и 144
0,007
0,656
F13B_GDTYPAELYITGSILR_vs_LYAM1_SYYWIGIR
84 и 120
0,020
0,636
AFAM_HFQNLGK_vs_TENX_LNWEAPPGAFDSFLLR
38 и 141
0,034
0,623
F13B_GDTYPAELYITGSILR_vs_PSG3_VSAPSGTGHLPGLNPL
84 и 134
0,022
0,633
AFAM_HFQNLGK_vs_TENX_LSQLSVTDVTTSSLR
38 и 142
0,034
0,623
F13B_GDTYPAELYITGSILR_vs_TENX_LNWEAPPGAFDSFLLR
84 и 141
0,011
0,647
AFAM_HFQNLGK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
38 и 144
0,030
0,626
F13B_GDTYPAELYITGSILR_vs_TENX_LSQLSVTDVTTSSLR
84 и 142
0,006
0,658
ANGT_DPTFIPAPIQAK_vs_C163A_INPASLDK
42 и 54
0,032
0,625
F13B_GDTYPAELYITGSILR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
84 и 144
0,024
0,631
ANGT_DPTFIPAPIQAK_vs_CHL1_VIAVNEVGR
42 и 66
0,026
0,629
HABP2_FLNWIK_vs_C163A_INPASLDK
92 и 54
0,027
0,628
ANGT_DPTFIPAPIQAK_vs_CRIS3_AVSPPAR
42 и 78
0,043
0,618
HABP2_FLNWIK_vs_CHL1_VIAVNEVGR
92 и 66
0,040
0,620
ANGT_DPTFIPAPIQAK_vs_CRIS3_YEDLYSNCK
42 и 79
0,038
0,621
HABP2_FLNWIK_vs_FBLN1_TGYYFDGISR
92 и 86
0,028
0,628
ANGT_DPTFIPAPIQAK_vs_CSH_AHQLAIDTYQEFEETYIPK
42 и 80
0,006
0,658
HABP2_FLNWIK_vs_LYAM1_SYYWIGIR
92 и 120
0,022
0,633
ANGT_DPTFIPAPIQAK_vs_CSH_ISLLLIESWLEPVR
42 и 81
0,035
0,622
HABP2_FLNWIK_vs_PSG3_VSAPSGTGHLPGLNPL
92 и 134
0,016
0,640
ANGT_DPTFIPAPIQAK_vs_FBLN1_TGYYFDGISR
42 и 86
0,011
0,647
HABP2_FLNWIK_vs_SOM2.CSH_SVEGSCGF
92 и 139
0,048
0,617
ANGT_DPTFIPAPIQAK_vs_LYAM1_SYYWIGIR
42 и 120
0,012
0,645
HABP2_FLNWIK_vs_TENX_LNWEAPPGAFDSFLLR
92 и 141
0,014
0,642
ANGT_DPTFIPAPIQAK_vs_PGRP2_AGLLRPDYALLGHR
42 и 126
0,031
0,625
HABP2_FLNWIK_vs_TENX_LSQLSVTDVTTSSLR
92 и 142
0,012
0,646
ANGT_DPTFIPAPIQAK_vs_PSG1_FQLPGQK
42 и 131
0,048
0,615
HABP2_FLNWIK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
92 и 144
0,008
0,654
ANGT_DPTFIPAPIQAK_vs_PSG3_VSAPSGTGHLPGLNPL
42 и 134
0,006
0,660
HABP2_FLNWIK_vs_VTDB_ELPEHTVK
92 и 147
0,042
0,618
ANGT_DPTFIPAPIQAK_vs_SHBG_IALGGLLFPASNLR
42 и 18
0,012
0,646
HEMO_NFPSPVDAAFR_vs_LYAM1_SYYWIGIR
93 и 120
0,049
0,614
ANGT_DPTFIPAPIQAK_vs_SOM2.CSH_NYGLLYCFR
42 и 138
0,046
0,616
HEMO_NFPSPVDAAFR_vs_TENX_LNWEAPPGAFDSFLLR
93 и 141
0,017
0,639
ANGT_DPTFIPAPIQAK_vs_SOM2.CSH_SVEGSCGF
42 и 139
0,007
0,660
HEMO_NFPSPVDAAFR_vs_TENX_LSQLSVTDVTTSSLR
93 и 142
0,028
0,628
ANGT_DPTFIPAPIQAK_vs_SPRL1_VLTHSELAPLR
42 и 140
0,029
0,627
HEMO_NFPSPVDAAFR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
93 и 144
0,041
0,619
ANGT_DPTFIPAPIQAK_vs_TENX_LNWEAPPGAFDSFLLR
42 и 141
0,012
0,646
IBP4_QCHPALDGQR_vs_C163A_INPASLDK
2 и 54
0,031
0,625
ANGT_DPTFIPAPIQAK_vs_TENX_LSQLSVTDVTTSSLR
42 и 142
0,008
0,654
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,042
0,618
ANGT_DPTFIPAPIQAK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
42 и 144
0,006
0,660
IBP4_QCHPALDGQR_vs_CSH_AHQLAIDTYQEFEETYIPK
2 и 80
0,048
0,615
APOC3_GWVTDGFSSLK_vs_ALS_IRPHTFTGLSGLR
47 и 40
0,030
0,626
IBP4_QCHPALDGQR_vs_FBLN1_TGYYFDGISR
2 и 86
0,030
0,626
APOC3_GWVTDGFSSLK_vs_C163A_INPASLDK
47 и 54
0,001
0,689
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,019
0,636
APOC3_GWVTDGFSSLK_vs_CHL1_VIAVNEVGR
47 и 66
0,046
0,616
IBP4_QCHPALDGQR_vs_PGRP2_AGLLRPDYALLGHR
2 и 126
0,020
0,635
APOC3_GWVTDGFSSLK_vs_CRIS3_AVSPPAR
47 и 78
0,013
0,645
IBP4_QCHPALDGQR_vs_PSG3_VSAPSGTGHLPGLNPL
2 и 134
0,008
0,654
APOC3_GWVTDGFSSLK_vs_CRIS3_YEDLYSNCK
47 и 79
0,009
0,651
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,033
0,624
APOC3_GWVTDGFSSLK_vs_CSH_AHQLAIDTYQEFEETYIPK
47 и 80
0,028
0,628
IBP4_QCHPALDGQR_vs_SPRL1_VLTHSELAPLR
2 и 140
0,041
0,619
APOC3_GWVTDGFSSLK_vs_FBLN1_TGYYFDGISR
47 и 86
0,038
0,620
IBP4_QCHPALDGQR_vs_TENX_LNWEAPPGAFDSFLLR
2 и 141
0,013
0,645
APOC3_GWVTDGFSSLK_vs_IBP3_FLNVLSPR
47 и 99
0,020
0,635
IBP4_QCHPALDGQR_vs_TENX_LSQLSVTDVTTSSLR
2 и 142
0,011
0,648
APOC3_GWVTDGFSSLK_vs_IBP3_YGQPLPGYTTK
47 и 100
0,044
0,617
IBP4_QCHPALDGQR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
2 и 144
0,007
0,656
APOC3_GWVTDGFSSLK_vs_IGF2_GIVEECCFR
47 и 103
0,029
0,627
INHBC_LDFHFSSDR_vs_C163A_INPASLDK
107 и 54
0,011
0,647
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,011
0,648
INHBC_LDFHFSSDR_vs_FBLN1_TGYYFDGISR
107 и 86
0,038
0,620
APOC3_GWVTDGFSSLK_vs_PGRP2_AGLLRPDYALLGHR
47 и 126
0,037
0,621
INHBC_LDFHFSSDR_vs_IGF2_GIVEECCFR
107 и 103
0,047
0,615
APOC3_GWVTDGFSSLK_vs_PSG1_FQLPGQK
47 и 131
0,034
0,623
INHBC_LDFHFSSDR_vs_PGRP2_AGLLRPDYALLGHR
107 и 126
0,043
0,618
APOC3_GWVTDGFSSLK_vs_PSG3_VSAPSGTGHLPGLNPL
47 и 134
0,010
0,651
INHBC_LDFHFSSDR_vs_PSG3_VSAPSGTGHLPGLNPL
107 и 134
0,009
0,652
APOC3_GWVTDGFSSLK_vs_SHBG_IALGGLLFPASNLR
47 и 18
0,048
0,615
INHBC_LDFHFSSDR_vs_SPRL1_VLTHSELAPLR
107 и 140
0,026
0,629
APOC3_GWVTDGFSSLK_vs_SOM2.CSH_SVEGSCGF
47 и 139
0,043
0,619
INHBC_LDFHFSSDR_vs_TENX_LNWEAPPGAFDSFLLR
107 и 141
0,004
0,666
APOC3_GWVTDGFSSLK_vs_TENX_LNWEAPPGAFDSFLLR
47 и 141
0,015
0,641
INHBC_LDFHFSSDR_vs_TENX_LSQLSVTDVTTSSLR
107 и 142
0,006
0,658
APOC3_GWVTDGFSSLK_vs_TENX_LSQLSVTDVTTSSLR
47 и 142
0,015
0,642
INHBC_LDFHFSSDR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
107 и 144
0,016
0,640
APOC3_GWVTDGFSSLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
47 и 144
0,012
0,645
ITIH4_NPLVWVHASPEHVVVTR_vs_CHL1_VIAVNEVGR
113 и 66
0,044
0,617
APOC3_GWVTDGFSSLK_vs_VTDB_ELPEHTVK
47 и 147
0,041
0,619
ITIH4_NPLVWVHASPEHVVVTR_vs_CRIS3_AVSPPAR
113 и 78
0,045
0,616
APOH_ATVVYQGER_vs_C163A_INPASLDK
48 и 54
0,032
0,625
ITIH4_NPLVWVHASPEHVVVTR_vs_CRIS3_YEDLYSNCK
113 и 79
0,035
0,622
APOH_ATVVYQGER_vs_CHL1_VIAVNEVGR
48 и 66
0,049
0,614
ITIH4_NPLVWVHASPEHVVVTR_vs_CSH_AHQLAIDTYQEFEETYIPK
113 и 80
0,042
0,618
APOH_ATVVYQGER_vs_CSH_AHQLAIDTYQEFEETYIPK
48 и 80
0,040
0,619
ITIH4_NPLVWVHASPEHVVVTR_vs_FBLN1_TGYYFDGISR
113 и 86
0,041
0,619
APOH_ATVVYQGER_vs_FBLN1_TGYYFDGISR
48 и 86
0,019
0,636
ITIH4_NPLVWVHASPEHVVVTR_vs_LYAM1_SYYWIGIR
113 и 120
0,017
0,638
APOH_ATVVYQGER_vs_LYAM1_SYYWIGIR
48 и 120
0,017
0,638
ITIH4_NPLVWVHASPEHVVVTR_vs_PGRP2_AGLLRPDYALLGHR
113 и 126
0,027
0,629
APOH_ATVVYQGER_vs_PGRP2_AGLLRPDYALLGHR
48 и 126
0,031
0,626
ITIH4_NPLVWVHASPEHVVVTR_vs_PSG3_VSAPSGTGHLPGLNPL
113 и 134
0,042
0,618
APOH_ATVVYQGER_vs_PSG3_VSAPSGTGHLPGLNPL
48 и 134
0,013
0,644
ITIH4_NPLVWVHASPEHVVVTR_vs_SHBG_IALGGLLFPASNLR
113 и 18
0,042
0,618
APOH_ATVVYQGER_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
48 и 135
0,024
0,631
ITIH4_NPLVWVHASPEHVVVTR_vs_SOM2.CSH_SVEGSCGF
113 и 139
0,045
0,618
APOH_ATVVYQGER_vs_SPRL1_VLTHSELAPLR
48 и 140
0,039
0,620
ITIH4_NPLVWVHASPEHVVVTR_vs_TENX_LNWEAPPGAFDSFLLR
113 и 141
0,016
0,641
APOH_ATVVYQGER_vs_TENX_LNWEAPPGAFDSFLLR
48 и 141
0,011
0,648
ITIH4_NPLVWVHASPEHVVVTR_vs_TENX_LSQLSVTDVTTSSLR
113 и 142
0,011
0,647
APOH_ATVVYQGER_vs_TENX_LSQLSVTDVTTSSLR
48 и 142
0,008
0,653
ITIH4_NPLVWVHASPEHVVVTR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
113 и 144
0,044
0,617
APOH_ATVVYQGER_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
48 и 144
0,015
0,642
KNG1_DIPTNSPELEETLTHTITK_vs_LYAM1_SYYWIGIR
116 и 120
0,042
0,618
C1QB_VPGLYYFTYHASSR_vs_ALS_IRPHTFTGLSGLR
55 и 40
0,030
0,626
KNG1_DIPTNSPELEETLTHTITK_vs_PSG3_VSAPSGTGHLPGLNPL
116 и 134
0,049
0,614
C1QB_VPGLYYFTYHASSR_vs_C163A_INPASLDK
55 и 54
0,010
0,650
KNG1_DIPTNSPELEETLTHTITK_vs_TENX_LNWEAPPGAFDSFLLR
116 и 141
0,038
0,621
C1QB_VPGLYYFTYHASSR_vs_CHL1_VIAVNEVGR
55 и 66
0,011
0,647
KNG1_DIPTNSPELEETLTHTITK_vs_TENX_LSQLSVTDVTTSSLR
116 и 142
0,024
0,631
C1QB_VPGLYYFTYHASSR_vs_CRIS3_AVSPPAR
55 и 78
0,017
0,638
KNG1_DIPTNSPELEETLTHTITK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
116 и 144
0,043
0,618
C1QB_VPGLYYFTYHASSR_vs_CRIS3_YEDLYSNCK
55 и 79
0,022
0,633
KNG1_QVVAGLNFR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
117 и 144
0,044
0,617
C1QB_VPGLYYFTYHASSR_vs_CSH_AHQLAIDTYQEFEETYIPK
55 и 80
0,004
0,669
LBP_ITGFLKPGK_vs_C163A_INPASLDK
118 и 54
0,037
0,621
C1QB_VPGLYYFTYHASSR_vs_CSH_ISLLLIESWLEPVR
55 и 81
0,010
0,650
LBP_ITGFLKPGK_vs_PSG3_VSAPSGTGHLPGLNPL
118 и 134
0,028
0,628
C1QB_VPGLYYFTYHASSR_vs_FBLN1_TGYYFDGISR
55 и 86
0,005
0,665
LBP_ITLPDFTGDLR_vs_C163A_INPASLDK
119 и 54
0,021
0,634
C1QB_VPGLYYFTYHASSR_vs_IBP2_LIQGAPTIR
55 и 98
0,036
0,622
LBP_ITLPDFTGDLR_vs_LYAM1_SYYWIGIR
119 и 120
0,045
0,617
C1QB_VPGLYYFTYHASSR_vs_IBP3_FLNVLSPR
55 и 99
0,022
0,633
LBP_ITLPDFTGDLR_vs_PSG3_VSAPSGTGHLPGLNPL
119 и 134
0,017
0,639
C1QB_VPGLYYFTYHASSR_vs_IBP3_YGQPLPGYTTK
55 и 100
0,027
0,628
PEDF_LQSLFDSPDFSK_vs_C163A_INPASLDK
124 и 54
0,042
0,618
C1QB_VPGLYYFTYHASSR_vs_IGF2_GIVEECCFR
55 и 103
0,011
0,648
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,034
0,623
C1QB_VPGLYYFTYHASSR_vs_ITIH4_ILDDLSPR
55 и 112
0,024
0,631
PEDF_LQSLFDSPDFSK_vs_PSG3_VSAPSGTGHLPGLNPL
124 и 134
0,044
0,617
C1QB_VPGLYYFTYHASSR_vs_LYAM1_SYYWIGIR
55 и 120
0,004
0,668
PEDF_LQSLFDSPDFSK_vs_TENX_LNWEAPPGAFDSFLLR
124 и 141
0,019
0,636
C1QB_VPGLYYFTYHASSR_vs_NCAM1_GLGEISAASEFK
55 и 121
0,019
0,636
PEDF_LQSLFDSPDFSK_vs_TENX_LSQLSVTDVTTSSLR
124 и 142
0,009
0,651
C1QB_VPGLYYFTYHASSR_vs_PGRP2_AGLLRPDYALLGHR
55 и 126
0,004
0,669
PEDF_TVQAVLTVPK_vs_C163A_INPASLDK
125 и 54
0,009
0,652
C1QB_VPGLYYFTYHASSR_vs_PSG1_FQLPGQK
55 и 131
0,020
0,636
PEDF_TVQAVLTVPK_vs_CHL1_VIAVNEVGR
125 и 66
0,025
0,630
C1QB_VPGLYYFTYHASSR_vs_PSG3_VSAPSGTGHLPGLNPL
55 и 134
0,004
0,665
PEDF_TVQAVLTVPK_vs_CRIS3_AVSPPAR
125 и 78
0,049
0,614
C1QB_VPGLYYFTYHASSR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
55 и 135
0,024
0,632
PEDF_TVQAVLTVPK_vs_CRIS3_YEDLYSNCK
125 и 79
0,044
0,617
C1QB_VPGLYYFTYHASSR_vs_SHBG_IALGGLLFPASNLR
55 и 18
0,003
0,673
PEDF_TVQAVLTVPK_vs_CSH_AHQLAIDTYQEFEETYIPK
125 и 80
0,039
0,620
C1QB_VPGLYYFTYHASSR_vs_SOM2.CSH_NYGLLYCFR
55 и 138
0,008
0,654
PEDF_TVQAVLTVPK_vs_FBLN1_TGYYFDGISR
125 и 86
0,041
0,619
C1QB_VPGLYYFTYHASSR_vs_SOM2.CSH_SVEGSCGF
55 и 139
0,010
0,652
PEDF_TVQAVLTVPK_vs_LYAM1_SYYWIGIR
125 и 120
0,012
0,646
C1QB_VPGLYYFTYHASSR_vs_SPRL1_VLTHSELAPLR
55 и 140
0,010
0,651
PEDF_TVQAVLTVPK_vs_PSG3_VSAPSGTGHLPGLNPL
125 и 134
0,011
0,647
C1QB_VPGLYYFTYHASSR_vs_TENX_LNWEAPPGAFDSFLLR
55 и 141
0,004
0,666
PEDF_TVQAVLTVPK_vs_SOM2.CSH_SVEGSCGF
125 и 139
0,040
0,621
C1QB_VPGLYYFTYHASSR_vs_TENX_LSQLSVTDVTTSSLR
55 и 142
0,002
0,681
PEDF_TVQAVLTVPK_vs_TENX_LNWEAPPGAFDSFLLR
125 и 141
0,005
0,663
C1QB_VPGLYYFTYHASSR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
55 и 144
0,004
0,666
PEDF_TVQAVLTVPK_vs_TENX_LSQLSVTDVTTSSLR
125 и 142
0,003
0,673
C1QB_VPGLYYFTYHASSR_vs_VTDB_ELPEHTVK
55 и 147
0,034
0,623
PEDF_TVQAVLTVPK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
125 и 144
0,006
0,658
CAH1_GGPFSDSYR_vs_C163A_INPASLDK
56 и 54
0,033
0,624
PRDX2_GLFIIDGK_vs_C163A_INPASLDK
128 и 54
0,013
0,644
CAH1_GGPFSDSYR_vs_CRIS3_YEDLYSNCK
56 и 79
0,045
0,617
PRDX2_GLFIIDGK_vs_CRIS3_AVSPPAR
128 и 78
0,038
0,621
CAH1_GGPFSDSYR_vs_PSG1_FQLP GQK
56 и 131
0,046
0,616
PRDX2_GLFIIDGK_vs_CRIS3_YEDLYSNCK
128 и 79
0,024
0,632
CAH1_GGPFSDSYR_vs_PSG3_VSAPSGTGHLPGLNPL
56 и 134
0,029
0,627
PRDX2_GLFIIDGK_vs_LYAM1_SYYWIGIR
128 и 120
0,035
0,623
CAH1_GGPFSDSYR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
56 и 135
0,048
0,615
PRDX2_GLFIIDGK_vs_PSG3_VSAPSGTGHLPGLNPL
128 и 134
0,020
0,635
CAH1_GGPFSDSYR_vs_SHBG_IALGGLLFPASNLR
56 и 18
0,044
0,617
PRDX2_GLFIIDGK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
128 и 135
0,047
0,615
CAH1_GGPFSDSYR_vs_TENX_LNWEAPPGAFDSFLLR
56 и 141
0,038
0,621
PRDX2_GLFIIDGK_vs_SHBG_IALGGLLFPASNLR
128 и 18
0,029
0,627
CAH1_GGPFSDSYR_vs_TENX_LSQLSVTDVTTSSLR
56 и 142
0,026
0,630
PRDX2_GLFIIDGK_vs_TENX_LNWEAPPGAFDSFLLR
128 и 141
0,022
0,633
CAH1_GGPFSDSYR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
56 и 144
0,040
0,619
PRDX2_GLFIIDGK_vs_TENX_LSQLSVTDVTTSSLR
128 и 142
0,021
0,634
CBPN_EALIQFLEQVHQGIK_vs_FBLN1_TGYYFDGISR
59 и 86
0,050
0,614
PRDX2_GLFIIDGK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
128 и 144
0,030
0,626
CBPN_EALIQFLEQVHQGIK_vs_LYAM1_SYYWIGIR
59 и 120
0,030
0,626
PSG2_IHPSYTNYR_vs_FBLN1_TGYYFDGISR
133 и 86
0,017
0,639
CBPN_EALIQFLEQVHQGIK_vs_TENX_LNWEAPPGAFDSFLLR
59 и 141
0,028
0,627
PSG2_IHPSYTNYR_vs_LYAM1_SYYWIGIR
133 и 120
0,037
0,621
CBPN_EALIQFLEQVHQGIK_vs_TENX_LSQLSVTDVTTSSLR
59 и 142
0,029
0,627
PSG2_IHPSYTNYR_vs_PSG3_VSAPSGTGHLPGLNPL
133 и 134
0,042
0,618
CBPN_EALIQFLEQVHQGIK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
59 и 144
0,049
0,614
PSG2_IHPSYTNYR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
133 и 135
0,016
0,640
CBPN_NNANGVDLNR_vs_FBLN1_TGYYFDGISR
60 и 86
0,045
0,617
PSG2_IHPSYTNYR_vs_TENX_LNWEAPPGAFDSFLLR
133 и 141
0,041
0,619
CBPN_NNANGVDLNR_vs_TENX_LNWEAPPGAFDSFLLR
60 и 141
0,045
0,617
PSG2_IHPSYTNYR_vs_TENX_LSQLSVTDVTTSSLR
133 и 142
0,026
0,629
CD14_LTVGAAQVPAQLLVGALR_vs_C163A_INPASLDK
61 и 54
0,033
0,624
PTGDS_GPGEDFR_vs_PSG3_VSAPSGTGHLPGLNPL
137 и 134
0,048
0,615
CD14_LTVGAAQVPAQLLVGALR_vs_FBLN1_TGYYFDGISR
61 и 86
0,036
0,622
PTGDS_GPGEDFR_vs_TENX_LNWEAPPGAFDSFLLR
137 и 141
0,036
0,622
CD14_LTVGAAQVPAQLLVGALR_vs_LYAM1_SYYWIGIR
61 и 120
0,011
0,648
PTGDS_GPGEDFR_vs_TENX_LSQLSVTDVTTSSLR
137 и 142
0,043
0,618
CD14_LTVGAAQVPAQLLVGALR_vs_PGRP2_AGLLRPDYALLGHR
61 и 126
0,032
0,625
PTGDS_GPGEDFR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
137 и 144
0,028
0,628
CD14_LTVGAAQVPAQLLVGALR_vs_PSG3_VSAPSGTGHLPGLNPL
61 и 134
0,024
0,631
VTNC_GQYCYELDEK_vs_C163A_INPASLDK
149 и 54
0,005
0,664
CD14_LTVGAAQVPAQLLVGALR_vs_SPRL1_VLTHSELAPLR
61 и 140
0,041
0,619
VTNC_GQYCYELDEK_vs_CHL1_VIAVNEVGR
149 и 66
0,019
0,636
CD14_LTVGAAQVPAQLLVGALR_vs_TENX_LNWEAPPGAFDSFLLR
61 и 141
0,007
0,657
VTNC_GQYCYELDEK_vs_CRIS3_AVSPPAR
149 и 78
0,021
0,634
CD14_LTVGAAQVPAQLLVGALR_vs_TENX_LSQLSVTDVTTSSLR
61 и 142
0,004
0,665
VTNC_GQYCYELDEK_vs_CRIS3_YEDLYSNCK
149 и 79
0,020
0,635
CD14_LTVGAAQVPAQLLVGALR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
61 и 144
0,007
0,657
VTNC_GQYCYELDEK_vs_CSH_AHQLAIDTYQEFEETYIPK
149 и 80
0,027
0,629
CD14_SWLAELQQWLKPGLK_vs_C163A_INPASLDK
62 и 54
0,037
0,621
VTNC_GQYCYELDEK_vs_FBLN1_TGYYFDGISR
149 и 86
0,012
0,645
CD14_SWLAELQQWLKPGLK_vs_FBLN1_TGYYFDGISR
62 и 86
0,034
0,623
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,002
0,678
CD14_SWLAELQQWLKPGLK_vs_LYAM1_SYYWIGIR
62 и 120
0,026
0,630
VTNC_GQYCYELDEK_vs_NCAM1_GLGEISAASEFK
149 и 121
0,039
0,620
CD14_SWLAELQQWLKPGLK_vs_PSG3_VSAPSGTGHLPGLNPL
62 и 134
0,043
0,618
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,006
0,659
CD14_SWLAELQQWLKPGLK_vs_TENX_LNWEAPPGAFDSFLLR
62 и 141
0,010
0,649
VTNC_GQYCYELDEK_vs_PSG3_VSAPSGTGHLPGLNPL
149 и 134
0,004
0,666
CD14_SWLAELQQWLKPGLK_vs_TENX_LSQLSVTDVTTSSLR
62 и 142
0,006
0,661
VTNC_GQYCYELDEK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
149 и 135
0,037
0,621
CD14_SWLAELQQWLKPGLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
62 и 144
0,012
0,646
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,013
0,644
CFAB_YGLVTYATYPK_vs_C163A_INPASLDK
64 и 54
0,025
0,630
VTNC_GQYCYELDEK_vs_SOM2.CSH_SVEGSCGF
149 и 139
0,020
0,637
CFAB_YGLVTYATYPK_vs_PSG3_VSAPSGTGHLPGLNPL
64 и 134
0,040
0,620
VTNC_GQYCYELDEK_vs_SPRL1_VLTHSELAPLR
149 и 140
0,023
0,632
CFAB_YGLVTYATYPK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
64 и 144
0,011
0,648
VTNC_GQYCYELDEK_vs_TENX_LNWEAPPGAFDSFLLR
149 и 141
0,008
0,653
CLUS_ASSIIDELFQDR_vs_LYAM1_SYYWIGIR
67 и 120
0,043
0,618
VTNC_GQYCYELDEK_vs_TENX_LSQLSVTDVTTSSLR
149 и 142
0,007
0,657
CLUS_ASSIIDELFQDR_vs_TENX_LNWEAPPGAFDSFLLR
67 и 141
0,030
0,626
VTNC_GQYCYELDEK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
149 и 144
0,001
0,690
CLUS_ASSIIDELFQDR_vs_TENX_LSQLSVTDVTTSSLR
67 и 142
0,023
0,632
VTNC_GQYCYELDEK_vs_VTDB_ELPEHTVK
149 и 147
0,020
0,636
CLUS_LFDSDPITVTVPVEVSR_vs_TENX_LNWEAPPGAFDSFLLR
68 и 141
0,043
0,618
VTNC_VDTVDPPYPR_vs_C163A_INPASLDK
150 и 54
0,018
0,638
CLUS_LFDSDPITVTVPVEVSR_vs_TENX_LSQLSVTDVTTSSLR
68 и 142
0,041
0,619
VTNC_VDTVDPPYPR_vs_CRIS3_AVSPPAR
150 и 78
0,040
0,620
CO5_TLLPVSKPEIR_vs_FBLN1_TGYYFDGISR
70 и 86
0,045
0,617
VTNC_VDTVDPPYPR_vs_CRIS3_YEDLYSNCK
150 и 79
0,038
0,621
CO5_TLLPVSKPEIR_vs_LYAM1_SYYWIGIR
70 и 120
0,014
0,643
VTNC_VDTVDPPYPR_vs_FBLN1_TGYYFDGISR
150 и 86
0,040
0,619
CO5_TLLPVSKPEIR_vs_PGRP2_AGLLRPDYALLGHR
70 и 126
0,043
0,618
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,004
0,666
CO5_TLLPVSKPEIR_vs_PSG3_VSAPSGTGHLPGLNPL
70 и 134
0,019
0,636
VTNC_VDTVDPPYPR_vs_PGRP2_AGLLRPDYALLGHR
150 и 126
0,027
0,629
CO5_TLLPVSKPEIR_vs_TENX_LNWEAPPGAFDSFLLR
70 и 141
0,016
0,640
VTNC_VDTVDPPYPR_vs_PSG3_VSAPSGTGHLPGLNPL
150 и 134
0,007
0,658
CO5_TLLPVSKPEIR_vs_TENX_LSQLSVTDVTTSSLR
70 и 142
0,011
0,647
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,030
0,626
CO5_TLLPVSKPEIR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
70 и 144
0,010
0,650
VTNC_VDTVDPPYPR_vs_SOM2.CSH_SVEGSCGF
150 и 139
0,046
0,617
CO5_VFQFLEK_vs_LYAM1_SYYWIGIR
71 и 120
0,027
0,628
VTNC_VDTVDPPYPR_vs_SPRL1_VLTHSELAPLR
150 и 140
0,043
0,618
CO5_VFQFLEK_vs_TENX_LNWEAPPGAFDSFLLR
71 и 141
0,016
0,641
VTNC_VDTVDPPYPR_vs_TENX_LNWEAPPGAFDSFLLR
150 и 141
0,021
0,634
CO5_VFQFLEK_vs_TENX_LSQLSVTDVTTSSLR
71 и 142
0,020
0,635
VTNC_VDTVDPPYPR_vs_TENX_LSQLSVTDVTTSSLR
150 и 142
0,016
0,640
CO5_VFQFLEK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
71 и 144
0,016
0,640
VTNC_VDTVDPPYPR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
150 и 144
0,004
0,669
CO6_ALNHLPLEYNSALYSR_vs_TENX_LNWEAPPGAFDSFLLR
72 и 141
0,019
0,637
VTNC_VDTVDPPYPR_vs_VTDB_ELPEHTVK
150 и 147
0,032
0,625
Таблица 33: эффективность классификации по обращениям, недели 18 и 19.
AUROC обращения для недель беременности 18 и 19 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, без BMI-стратификации.
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
ANGT_DPTFIPAPIQAK_vs_IGF2_GIVEECCFR
42 и 103
0,039
0,650
F13B_GDTYPAELYITGSILR_vs_IGF2_GIVEECCFR
84 и 103
0,017
0,674
ANGT_DPTFIPAPIQAK_vs_LYAM1_SYYWIGIR
42 и 120
0,023
0,665
F13B_GDTYPAELYITGSILR_vs_LYAM1_SYYWIGIR
84 и 120
0,006
0,700
APOC3_GWVTDGFSSLK_vs_C163A_INPASLDK
47 и 54
0,018
0,673
FETUA_FSVVYAK_vs_PSG9_LFIPQITR
88 и 136
0,030
0,658
APOC3_GWVTDGFSSLK_vs_CRIS3_YEDLYSNCK
47 и 79
0,040
0,650
FETUA_HTLNQIDEVK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
89 и 135
0,037
0,652
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,019
0,671
FETUA_HTLNQIDEVK_vs_PSG9_LFIPQITR
89 и 136
0,017
0,673
APOH_ATVVYQGER_vs_CRIS3_YEDLYSNCK
48 и 79
0,042
0,648
HABP2_FLNWIK_vs_C163A_INPASLDK
92 и 54
0,021
0,668
APOH_ATVVYQGER_vs_IGF2_GIVEECCFR
48 и 103
0,049
0,644
HABP2_FLNWIK_vs_IGF2_GIVEECCFR
92 и 103
0,030
0,658
APOH_ATVVYQGER_vs_LYAM1_SYYWIGIR
48 и 120
0,017
0,675
HABP2_FLNWIK_vs_LYAM1_SYYWIGIR
92 и 120
0,015
0,677
B2MG_VNHVTLSQPK_vs_C163A_INPASLDK
51 и 54
0,026
0,663
HABP2_FLNWIK_vs_TENX_LNWEAPPGAFDSFLLR
92 и 141
0,049
0,644
B2MG_VNHVTLSQPK_vs_LYAM1_SYYWIGIR
51 и 120
0,019
0,671
HABP2_FLNWIK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
92 и 144
0,021
0,668
CATD_VGFAEAAR_vs_C163A_INPASLDK
57 и 54
0,029
0,659
HABP2_FLNWIK_vs_VTDB_ELPEHTVK
92 и 147
0,037
0,652
CBPN_EALIQFLEQVHQGIK_vs_PSG9_LFIPQITR
59 и 136
0,032
0,656
HEMO_NFPSPVDAAFR_vs_IGF2_GIVEECCFR
93 и 103
0,045
0,647
CBPN_NNANGVDLNR_vs_LYAM1_SYYWIGIR
60 и 120
0,038
0,652
HEMO_NFPSPVDAAFR_vs_LYAM1_SYYWIGIR
93 и 120
0,035
0,654
CD14_LTVGAAQVPAQLLVGALR_vs_C163A_INPASLDK
61 и 54
0,029
0,659
IBP4_QCHPALDGQR_vs_C163A_INPASLDK
2 и 54
0,029
0,660
CD14_LTVGAAQVPAQLLVGALR_vs_CSH_AHQLAIDTYQEFEETYIPK
61 и 80
0,039
0,651
IBP4_QCHPALDGQR_vs_CRIS3_AVSPPAR
2 и 78
0,040
0,649
CD14_LTVGAAQVPAQLLVGALR_vs_IGF2_GIVEECCFR
61 и 103
0,024
0,664
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,020
0,670
CD14_LTVGAAQVPAQLLVGALR_vs_LYAM1_SYYWIGIR
61 и 120
0,005
0,703
IBP4_QCHPALDGQR_vs_IGF2_GIVEECCFR
2 и 103
0,026
0,662
CD14_LTVGAAQVPAQLLVGALR_vs_TENX_LSQLSVTDVTTSSLR
61 и 142
0,032
0,657
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,005
0,705
CD14_LTVGAAQVPAQLLVGALR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
61 и 144
0,012
0,684
IBP6_GAQTLYVPNCDHR_vs_C163A_INPASLDK
101 и 54
0,015
0,678
CD14_LTVGAAQVPAQLLVGALR_vs_VTDB_ELPEHTVK
61 и 147
0,026
0,663
IBP6_GAQTLYVPNCDHR_vs_IGF2_GIVEECCFR
101 и 103
0,035
0,654
CD14_SWLAELQQWLKPGLK_vs_C163A_INPASLDK
62 и 54
0,036
0,653
IBP6_GAQTLYVPNCDHR_vs_LYAM1_SYYWIGIR
101 и 120
0,025
0,664
CD14_SWLAELQQWLKPGLK_vs_IGF2_GIVEECCFR
62 и 103
0,047
0,645
IBP6_HLDSVLQQLQTEVYR_vs_C163A_INPASLDK
102 и 54
0,015
0,678
CD14_SWLAELQQWLKPGLK_vs_LYAM1_SYYWIGIR
62 и 120
0,018
0,672
IBP6_HLDSVLQQLQTEVYR_vs_LYAM1_SYYWIGIR
102 и 120
0,032
0,656
CD14_SWLAELQQWLKPGLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
62 и 144
0,022
0,667
KNG1_DIPTNSPELEETLTHTITK_vs_IGF2_GIVEECCFR
116 и 103
0,048
0,644
CLUS_ASSIIDELFQDR_vs_C163A_INPASLDK
67 и 54
0,032
0,656
KNG1_DIPTNSPELEETLTHTITK_vs_LYAM1_SYYWIGIR
116 и 120
0,016
0,675
CLUS_ASSIIDELFQDR_vs_IBP3_FLNVLSPR
67 и 99
0,038
0,651
KNG1_QVVAGLNFR_vs_C163A_INPASLDK
117 и 54
0,049
0,643
CLUS_ASSIIDELFQDR_vs_IBP3_YGQPLPGYTTK
67 и 100
0,026
0,662
KNG1_QVVAGLNFR_vs_CRIS3_YEDLYSNCK
117 и 79
0,040
0,650
CLUS_ASSIIDELFQDR_vs_IGF2_GIVEECCFR
67 и 103
0,003
0,718
KNG1_QVVAGLNFR_vs_IGF2_GIVEECCFR
117 и 103
0,010
0,687
CLUS_ASSIIDELFQDR_vs_LYAM1_SYYWIGIR
67 и 120
0,004
0,712
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,007
0,697
CLUS_ASSIIDELFQDR_vs_VTDB_ELPEHTVK
67 и 147
0,029
0,659
KNG1_QVVAGLNFR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
117 и 144
0,043
0,648
CLUS_LFDSDPITVTVPVEVSR_vs_C163A_INPASLDK
68 и 54
0,029
0,660
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,046
0,646
CLUS_LFDSDPITVTVPVEVSR_vs_CRIS3_YEDLYSNCK
68 и 79
0,023
0,666
PEDF_TVQAVLTVPK_vs_LYAM1_SYYWIGIR
125 и 120
0,021
0,668
CLUS_LFDSDPITVTVPVEVSR_vs_CSH_AHQLAIDTYQEFEETYIPK
68 и 80
0,047
0,645
PSG2_IHPSYTNYR_vs_C163A_INPASLDK
133 и 54
0,024
0,665
CLUS_LFDSDPITVTVPVEVSR_vs_IBP3_FLNVLSPR
68 и 99
0,017
0,674
PSG2_IHPSYTNYR_vs_CRIS3_AVSPPAR
133 и 78
0,021
0,668
CLUS_LFDSDPITVTVPVEVSR_vs_IBP3_YGQPLPGYTTK
68 и 100
0,011
0,686
PSG2_IHPSYTNYR_vs_CRIS3_YEDLYSNCK
133 и 79
0,013
0,681
CLUS_LFDSDPITVTVPVEVSR_vs_IGF2_GIVEECCFR
68 и 103
0,001
0,734
PSG2_IHPSYTNYR_vs_CSH_AHQLAIDTYQEFEETYIPK
133 и 80
0,020
0,670
CLUS_LFDSDPITVTVPVEVSR_vs_ITIH4_ILDDLSPR
68 и 112
0,048
0,644
PSG2_IHPSYTNYR_vs_CSH_ISLLLIESWLEPVR
133 и 81
0,041
0,649
CLUS_LFDSDPITVTVPVEVSR_vs_LYAM1_SYYWIGIR
68 и 120
0,003
0,713
PSG2_IHPSYTNYR_vs_FBLN1_TGYYFDGISR
133 и 86
0,010
0,688
CLUS_LFDSDPITVTVPVEVSR_vs_TENX_LNWEAPPGAFDSFLLR
68 и 141
0,030
0,658
PSG2_IHPSYTNYR_vs_IBP3_YGQPLPGYTTK
133 и 100
0,041
0,649
CLUS_LFDSDPITVTVPVEVSR_vs_TENX_LSQLSVTDVTTSSLR
68 и 142
0,047
0,645
PSG2_IHPSYTNYR_vs_IGF2_GIVEECCFR
133 и 103
0,030
0,659
CLUS_LFDSDPITVTVPVEVSR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
68 и 144
0,041
0,649
PSG2_IHPSYTNYR_vs_LYAM1_SYYWIGIR
133 и 120
0,007
0,696
CLUS_LFDSDPITVTVPVEVSR_vs_VTDB_ELPEHTVK
68 и 147
0,013
0,682
PSG2_IHPSYTNYR_vs_NCAM1_GLGEISAASEFK
133 и 121
0,050
0,643
CO5_TLLPVSKPEIR_vs_LYAM1_SYYWIGIR
70 и 120
0,021
0,668
PSG2_IHPSYTNYR_vs_PSG3_VSAPSGTGHLPGLNPL
133 и 134
0,024
0,664
CO5_VFQFLEK_vs_LYAM1_SYYWIGIR
71 и 120
0,027
0,661
PSG2_IHPSYTNYR_vs_SHBG_IALGGLLFPASNLR
133 и 18
0,033
0,655
CO6_ALNHLPLEYNSALYSR_vs_IGF2_GIVEECCFR
72 и 103
0,020
0,669
PSG2_IHPSYTNYR_vs_SOM2.CSH_NYGLLYCFR
133 и 138
0,037
0,652
CO6_ALNHLPLEYNSALYSR_vs_LYAM1_SYYWIGIR
72 и 120
0,009
0,689
PSG2_IHPSYTNYR_vs_SPRL1_VLTHSELAPLR
133 и 140
0,043
0,648
CO6_ALNHLPLEYNSALYSR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
72 и 144
0,021
0,669
PSG2_IHPSYTNYR_vs_TENX_LNWEAPPGAFDSFLLR
133 и 141
0,030
0,658
CO8A_SLLQPNK_vs_IGF2_GIVEECCFR
74 и 103
0,048
0,644
PSG2_IHPSYTNYR_vs_TENX_LSQLSVTDVTTSSLR
133 и 142
0,023
0,665
CO8A_SLLQPNK_vs_LYAM1_SYYWIGIR
74 и 120
0,010
0,687
PSG2_IHPSYTNYR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
133 и 144
0,037
0,652
CO8A_SLLQPNK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
74 и 144
0,028
0,660
PTGDS_GPGEDFR_vs_LYAM1_SYYWIGIR
137 и 120
0,045
0,646
CO8B_QALEEFQK_vs_LYAM1_SYYWIGIR
76 и 120
0,043
0,647
THBG_AVLHIGEK_vs_LYAM1_SYYWIGIR
143 и 120
0,036
0,653
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
82 и 135
0,050
0,643
VTNC_GQYCYELDEK_vs_C163A_INPASLDK
149 и 54
0,041
0,649
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PSG9_LFIPQITR
82 и 136
0,027
0,662
VTNC_GQYCYELDEK_vs_IGF2_GIVEECCFR
149 и 103
0,032
0,657
ENPP2_TYLHTYESEI_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
83 и 135
0,050
0,643
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,006
0,700
ENPP2_TYLHTYESEI_vs_PSG9_LFIPQITR
83 и 136
0,028
0,660
VTNC_GQYCYELDEK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
149 и 144
0,047
0,645
F13B_GDTYPAELYITGSILR_vs_CRIS3_YEDLYSNCK
84 и 79
0,045
0,646
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,015
0,678
Таблица 34: эффективность классификации по обращениям, недели 18 и 19.
AUROC обращения для недель беременности 18 и 19 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, с BMI-стратификацией (>22≤37).
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
A2GL_DLLLPQPDLR_vs_LYAM1_SYYWIGIR
34 и 120
0,040
0,686
KNG1_DIPTNSPELEETLTHTITK_vs_IGF2_GIVEECCFR
116 и 103
0,020
0,710
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,022
0,707
KNG1_DIPTNSPELEETLTHTITK_vs_LYAM1_SYYWIGIR
116 и 120
0,010
0,734
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,045
0,682
KNG1_QVVAGLNFR_vs_IBP3_FLNVLSPR
117 и 99
0,029
0,698
ANGT_DPTFIPAPIQAK_vs_IGF2_GIVEECCFR
42 и 103
0,029
0,699
KNG1_QVVAGLNFR_vs_IBP3_YGQPLPGYTTK
117 и 100
0,034
0,692
ANGT_DPTFIPAPIQAK_vs_LYAM1_SYYWIGIR
42 и 120
0,016
0,718
KNG1_QVVAGLNFR_vs_IGF2_GIVEECCFR
117 и 103
0,004
0,760
APOH_ATVVYQGER_vs_IGF2_GIVEECCFR
48 и 103
0,033
0,694
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,007
0,746
APOH_ATVVYQGER_vs_LYAM1_SYYWIGIR
48 и 120
0,026
0,702
LBP_ITGFLKPGK_vs_PSG9_LFIPQITR
118 и 136
0,044
0,682
B2MG_VNHVTLSQPK_vs_LYAM1_SYYWIGIR
51 и 120
0,018
0,715
PAPP1_DIPHWLNPTR_vs_LYAM1_SYYWIGIR
122 и 120
0,046
0,681
CD14_LTVGAAQVPAQLLVGALR_vs_IGF2_GIVEECCFR
61 и 103
0,028
0,700
PEDF_TVQAVLTVPK_vs_LYAM1_SYYWIGIR
125 и 120
0,018
0,714
CD14_LTVGAAQVPAQLLVGALR_vs_LYAM1_SYYWIGIR
61 и 120
0,006
0,748
PSG2_IHPSYTNYR_vs_ALS_IRPHTFTGLSGLR
133 и 40
0,038
0,688
CD14_LTVGAAQVPAQLLVGALR_vs_PGRP2_AGLLRPDYALLGHR
61 и 126
0,044
0,683
PSG2_IHPSYTNYR_vs_C163A_INPASLDK
133 и 54
0,012
0,728
CD14_LTVGAAQVPAQLLVGALR_vs_VTDB_ELPEHTVK
61 и 147
0,041
0,685
PSG2_IHPSYTNYR_vs_CHL1_VIAVNEVGR
133 и 66
0,036
0,691
CD14_SWLAELQQWLKPGLK_vs_LYAM1_SYYWIGIR
62 и 120
0,036
0,691
PSG2_IHPSYTNYR_vs_CRIS3_AVSPPAR
133 и 78
0,008
0,741
CLUS_ASSIIDELFQDR_vs_IGF2_GIVEECCFR
67 и 103
0,009
0,737
PSG2_IHPSYTNYR_vs_CRIS3_YEDLYSNCK
133 и 79
0,007
0,743
CLUS_ASSIIDELFQDR_vs_LYAM1_SYYWIGIR
67 и 120
0,010
0,733
PSG2_IHPSYTNYR_vs_CSH_AHQLAIDTYQEFEETYIPK
133 и 80
0,024
0,705
CLUS_LFDSDPITVTVPVEVSR_vs_IGF2_GIVEECCFR
68 и 103
0,021
0,710
PSG2_IHPSYTNYR_vs_CSH_ISLLLIESWLEPVR
133 и 81
0,044
0,683
CLUS_LFDSDPITVTVPVEVSR_vs_LYAM1_SYYWIGIR
68 и 120
0,027
0,700
PSG2_IHPSYTNYR_vs_FBLN1_TGYYFDGISR
133 и 86
0,017
0,716
CO5_TLLPVSKPEIR_vs_LYAM1_SYYWIGIR
70 и 120
0,017
0,717
PSG2_IHPSYTNYR_vs_IBP3_FLNVLSPR
133 и 99
0,019
0,712
CO5_VFQFLEK_vs_LYAM1_SYYWIGIR
71 и 120
0,035
0,691
PSG2_IHPSYTNYR_vs_IBP3_YGQPLPGYTTK
133 и 100
0,025
0,704
CO6_ALNHLPLEYNSALYSR_vs_IGF2_GIVEECCFR
72 и 103
0,016
0,719
PSG2_IHPSYTNYR_vs_IGF2_GIVEECCFR
133 и 103
0,010
0,733
CO6_ALNHLPLEYNSALYSR_vs_LYAM1_SYYWIGIR
72 и 120
0,014
0,724
PSG2_IHPSYTNYR_vs_ITIH4_ILDDLSPR
133 и 112
0,026
0,702
CO8A_SLLQPNK_vs_LYAM1_SYYWIGIR
74 и 120
0,024
0,705
PSG2_IHPSYTNYR_vs_LYAM1_SYYWIGIR
133 и 120
0,002
0,777
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
82 и 135
0,042
0,684
PSG2_IHPSYTNYR_vs_PGRP2_AGLLRPDYALLGHR
133 и 126
0,032
0,694
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PSG9_LFIPQITR
82 и 136
0,011
0,731
PSG2_IHPSYTNYR_vs_PSG3_VSAPSGTGHLPGLNPL
133 и 134
0,010
0,733
ENPP2_TYLHTYESEI_vs_NCAM1_GLGEISAASEFK
83 и 121
0,047
0,680
PSG2_IHPSYTNYR_vs_SHBG_IALGGLLFPASNLR
133 и 18
0,028
0,699
ENPP2_TYLHTYESEI_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
83 и 135
0,042
0,684
PSG2_IHPSYTNYR_vs_SOM2.CSH_NYGLLYCFR
133 и 138
0,036
0,690
ENPP2_TYLHTYESEI_vs_PSG9_LFIPQITR
83 и 136
0,012
0,727
PSG2_IHPSYTNYR_vs_SOM2.CSH_SVEGSCGF
133 и 139
0,045
0,682
ENPP2_TYLHTYESEI_vs_VTDB_ELPEHTVK
83 и 147
0,037
0,689
PSG2_IHPSYTNYR_vs_TENX_LNWEAPPGAFDSFLLR
133 и 141
0,024
0,705
F13B_GDTYPAELYITGSILR_vs_IGF2_GIVEECCFR
84 и 103
0,009
0,737
PSG2_IHPSYTNYR_vs_TENX_LSQLSVTDVTTSSLR
133 и 142
0,018
0,715
F13B_GDTYPAELYITGSILR_vs_LYAM1_SYYWIGIR
84 и 120
0,006
0,748
PSG2_IHPSYTNYR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
133 и 144
0,046
0,681
FETUA_FSVVYAK_vs_PSG9_LFIPQITR
88 и 136
0,043
0,684
PSG2_IHPSYTNYR_vs_VTDB_ELPEHTVK
133 и 147
0,021
0,710
FETUA_HTLNQIDEVK_vs_PSG9_LFIPQITR
89 и 136
0,037
0,689
PTGDS_GPGEDFR_vs_C163A_INPASLDK
137 и 54
0,036
0,690
HABP2_FLNWIK_vs_LYAM1_SYYWIGIR
92 и 120
0,045
0,682
PTGDS_GPGEDFR_vs_IGF2_GIVEECCFR
137 и 103
0,043
0,684
IBP4_QCHPALDGQR_vs_C163A_INPASLDK
2 и 54
0,048
0,679
PTGDS_GPGEDFR_vs_LYAM1_SYYWIGIR
137 и 120
0,031
0,696
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,043
0,684
VTNC_GQYCYELDEK_vs_IGF2_GIVEECCFR
149 и 103
0,022
0,707
IBP4_QCHPALDGQR_vs_IGF2_GIVEECCFR
2 и 103
0,010
0,733
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,006
0,749
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,004
0,759
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,023
0,705
Таблица 35: эффективность классификации по обращениям, недели 19 и 20.
AUROC обращения для недель беременности 19 и 20 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, без BMI-стратификации.
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
A2GL_DLLLPQPDLR_vs_PRG2_WNFAYWAAHQPWSR
34 и 129
0,018
0,618
ENPP2_TYLHTYESEI_vs_SHBG_IALGGLLFPASNLR
83 и 18
0,036
0,605
A2GL_DLLLPQPDLR_vs_PSG3_VSAPSGTGHLPGLNPL
34 и 134
0,013
0,624
FBLN3_IPSNPSHR_vs_SHBG_IALGGLLFPASNLR
87 и 18
0,038
0,604
A2GL_DLLLPQPDLR_vs_SHBG_IALGGLLFPASNLR
34 и 18
0,023
0,613
FETUA_FSVVYAK_vs_IGF2_GIVEECCFR
88 и 103
0,031
0,608
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,045
0,600
FETUA_FSVVYAK_vs_PRG2_WNFAYWAAHQPWSR
88 и 129
0,033
0,606
AFAM_HFQNLGK_vs_IBP3_YGQPLPGYTTK
38 и 100
0,021
0,616
FETUA_FSVVYAK_vs_PSG3_VSAPSGTGHLPGLNPL
88 и 134
0,017
0,619
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,013
0,624
FETUA_HTLNQIDEVK_vs_PRG2_WNFAYWAAHQPWSR
89 и 129
0,019
0,617
AFAM_HFQNLGK_vs_PRG2_WNFAYWAAHQPWSR
38 и 129
0,036
0,605
FETUA_HTLNQIDEVK_vs_PSG3_VSAPSGTGHLPGLNPL
89 и 134
0,010
0,628
AFAM_HFQNLGK_vs_PSG3_VSAPSGTGHLPGLNPL
38 и 134
0,031
0,608
FETUA_HTLNQIDEVK_vs_SHBG_IALGGLLFPASNLR
89 и 18
0,027
0,610
APOC3_GWVTDGFSSLK_vs_ALS_IRPHTFTGLSGLR
47 и 40
0,007
0,635
FETUA_HTLNQIDEVK_vs_TENX_LNWEAPPGAFDSFLLR
89 и 141
0,045
0,600
APOC3_GWVTDGFSSLK_vs_C163A_INPASLDK
47 и 54
0,009
0,631
HABP2_FLNWIK_vs_IGF2_GIVEECCFR
92 и 103
0,031
0,608
APOC3_GWVTDGFSSLK_vs_CHL1_VIAVNEVGR
47 и 66
0,027
0,611
HABP2_FLNWIK_vs_PSG3_VSAPSGTGHLPGLNPL
92 и 134
0,043
0,601
APOC3_GWVTDGFSSLK_vs_CRIS3_AVSPPAR
47 и 78
0,007
0,635
HEMO_NFPSPVDAAFR_vs_PSG3_VSAPSGTGHLPGLNPL
93 и 134
0,029
0,609
APOC3_GWVTDGFSSLK_vs_CRIS3_YEDLYSNCK
47 и 79
0,007
0,635
HEMO_NFPSPVDAAFR_vs_SHBG_IALGGLLFPASNLR
93 и 18
0,028
0,610
APOC3_GWVTDGFSSLK_vs_CSH_AHQLAIDTYQEFEETYIPK
47 и 80
0,015
0,621
IBP4_QCHPALDGQR_vs_ALS_IRPHTFTGLSGLR
2 и 40
0,024
0,612
APOC3_GWVTDGFSSLK_vs_CSH_ISLLLIESWLEPVR
47 и 81
0,027
0,610
IBP4_QCHPALDGQR_vs_CHL1_VIAVNEVGR
2 и 66
0,027
0,610
APOC3_GWVTDGFSSLK_vs_FBLN1_TGYYFDGISR
47 и 86
0,009
0,631
IBP4_QCHPALDGQR_vs_CRIS3_AVSPPAR
2 и 78
0,004
0,644
APOC3_GWVTDGFSSLK_vs_IBP2_LIQGAPTIR
47 и 98
0,037
0,604
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,003
0,650
APOC3_GWVTDGFSSLK_vs_IBP3_FLNVLSPR
47 и 99
0,001
0,659
IBP4_QCHPALDGQR_vs_FBLN1_TGYYFDGISR
2 и 86
0,020
0,617
APOC3_GWVTDGFSSLK_vs_IBP3_YGQPLPGYTTK
47 и 100
0,002
0,651
IBP4_QCHPALDGQR_vs_IBP3_FLNVLSPR
2 и 99
0,014
0,622
APOC3_GWVTDGFSSLK_vs_IGF2_GIVEECCFR
47 и 103
0,001
0,660
IBP4_QCHPALDGQR_vs_IBP3_YGQPLPGYTTK
2 и 100
0,005
0,639
APOC3_GWVTDGFSSLK_vs_ITIH4_ILDDLSPR
47 и 112
0,007
0,636
IBP4_QCHPALDGQR_vs_IGF2_GIVEECCFR
2 и 103
0,000
0,677
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,003
0,648
IBP4_QCHPALDGQR_vs_ITIH4_ILDDLSPR
2 и 112
0,003
0,650
APOC3_GWVTDGFSSLK_vs_NCAM1_GLGEISAASEFK
47 и 121
0,012
0,626
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,005
0,641
APOC3_GWVTDGFSSLK_vs_PGRP2_AGLLRPDYALLGHR
47 и 126
0,011
0,626
IBP4_QCHPALDGQR_vs_NCAM1_GLGEISAASEFK
2 и 121
0,027
0,610
APOC3_GWVTDGFSSLK_vs_PRG2_WNFAYWAAHQPWSR
47 и 129
0,004
0,642
IBP4_QCHPALDGQR_vs_PGRP2_AGLLRPDYALLGHR
2 и 126
0,011
0,627
APOC3_GWVTDGFSSLK_vs_PSG1_FQLPGQK
47 и 131
0,031
0,608
IBP4_QCHPALDGQR_vs_PRG2_WNFAYWAAHQPWSR
2 и 129
0,002
0,658
APOC3_GWVTDGFSSLK_vs_PSG3_VSAPSGTGHLPGLNPL
47 и 134
0,001
0,659
IBP4_QCHPALDGQR_vs_PSG3_VSAPSGTGHLPGLNPL
2 и 134
0,000
0,710
APOC3_GWVTDGFSSLK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
47 и 135
0,044
0,601
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,001
0,673
APOC3_GWVTDGFSSLK_vs_PSG9_LFIPQITR
47 и 136
0,042
0,602
IBP4_QCHPALDGQR_vs_SPRL1_VLTHSELAPLR
2 и 140
0,004
0,646
APOC3_GWVTDGFSSLK_vs_SHBG_IALGGLLFPASNLR
47 и 18
0,004
0,645
IBP4_QCHPALDGQR_vs_TENX_LNWEAPPGAFDSFLLR
2 и 141
0,002
0,655
APOC3_GWVTDGFSSLK_vs_SOM2.CSH_NYGLLYCFR
47 и 138
0,028
0,610
IBP4_QCHPALDGQR_vs_TENX_LSQLSVTDVTTSSLR
2 и 142
0,005
0,641
APOC3_GWVTDGFSSLK_vs_SOM2.CSH_SVEGSCGF
47 и 139
0,028
0,610
IBP4_QCHPALDGQR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
2 и 144
0,004
0,645
APOC3_GWVTDGFSSLK_vs_SPRL1_VLTHSELAPLR
47 и 140
0,008
0,634
IBP4_QCHPALDGQR_vs_VTDB_ELPEHTVK
2 и 147
0,002
0,658
APOC3_GWVTDGFSSLK_vs_TENX_LNWEAPPGAFDSFLLR
47 и 141
0,005
0,641
IBP6_GAQTLYVPNCDHR_vs_PRG2_WNFAYWAAHQPWSR
101 и 129
0,038
0,604
APOC3_GWVTDGFSSLK_vs_TENX_LSQLSVTDVTTSSLR
47 и 142
0,005
0,640
IBP6_GAQTLYVPNCDHR_vs_PSG3_VSAPSGTGHLPGLNPL
101 и 134
0,024
0,613
APOC3_GWVTDGFSSLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
47 и 144
0,006
0,638
IBP6_GAQTLYVPNCDHR_vs_SHBG_IALGGLLFPASNLR
101 и 18
0,045
0,600
APOC3_GWVTDGFSSLK_vs_VTDB_ELPEHTVK
47 и 147
0,005
0,641
IBP6_HLDSVLQQLQTEVYR_vs_PRG2_WNFAYWAAHQPWSR
102 и 129
0,032
0,607
APOH_ATVVYQGER_vs_CRIS3_YEDLYSNCK
48 и 79
0,043
0,601
IBP6_HLDSVLQQLQTEVYR_vs_PSG3_VSAPSGTGHLPGLNPL
102 и 134
0,023
0,613
APOH_ATVVYQGER_vs_IBP3_FLNVLSPR
48 и 99
0,031
0,608
IBP6_HLDSVLQQLQTEVYR_vs_SHBG_IALGGLLFPASNLR
102 и 18
0,032
0,607
APOH_ATVVYQGER_vs_IBP3_YGQPLPGYTTK
48 и 100
0,012
0,626
INHBC_LDFHFSSDR_vs_CRIS3_YEDLYSNCK
107 и 79
0,043
0,601
APOH_ATVVYQGER_vs_IGF2_GIVEECCFR
48 и 103
0,004
0,646
INHBC_LDFHFSSDR_vs_IBP3_FLNVLSPR
107 и 99
0,015
0,621
APOH_ATVVYQGER_vs_PRG2_WNFAYWAAHQPWSR
48 и 129
0,033
0,606
INHBC_LDFHFSSDR_vs_IBP3_YGQPLPGYTTK
107 и 100
0,011
0,627
APOH_ATVVYQGER_vs_PSG3_VSAPSGTGHLPGLNPL
48 и 134
0,003
0,650
INHBC_LDFHFSSDR_vs_IGF2_GIVEECCFR
107 и 103
0,002
0,652
APOH_ATVVYQGER_vs_SHBG_IALGGLLFPASNLR
48 и 18
0,014
0,622
INHBC_LDFHFSSDR_vs_ITIH4_ILDDLSPR
107 и 112
0,041
0,602
APOH_ATVVYQGER_vs_TENX_LNWEAPPGAFDSFLLR
48 и 141
0,025
0,612
INHBC_LDFHFSSDR_vs_LYAM1_SYYWIGIR
107 и 120
0,041
0,602
APOH_ATVVYQGER_vs_TENX_LSQLSVTDVTTSSLR
48 и 142
0,021
0,615
INHBC_LDFHFSSDR_vs_PGRP2_AGLLRPDYALLGHR
107 и 126
0,040
0,603
B2MG_VEHSDLSFSK_vs_PSG3_VSAPSGTGHLPGLNPL
50 и 134
0,036
0,605
INHBC_LDFHFSSDR_vs_PRG2_WNFAYWAAHQPWSR
107 и 129
0,016
0,620
B2MG_VNHVTLSQPK_vs_IGF2_GIVEECCFR
51 и 103
0,010
0,628
INHBC_LDFHFSSDR_vs_PSG3_VSAPSGTGHLPGLNPL
107 и 134
0,003
0,647
B2MG_VNHVTLSQPK_vs_PRG2_WNFAYWAAHQPWSR
51 и 129
0,020
0,617
INHBC_LDFHFSSDR_vs_SHBG_IALGGLLFPASNLR
107 и 18
0,008
0,632
B2MG_VNHVTLSQPK_vs_PSG3_VSAPSGTGHLPGLNPL
51 и 134
0,011
0,626
INHBC_LDFHFSSDR_vs_SPRL1_VLTHSELAPLR
107 и 140
0,035
0,605
B2MG_VNHVTLSQPK_vs_SHBG_IALGGLLFPASNLR
51 и 18
0,019
0,617
INHBC_LDFHFSSDR_vs_TENX_LNWEAPPGAFDSFLLR
107 и 141
0,007
0,634
B2MG_VNHVTLSQPK_vs_TENX_LSQLSVTDVTTSSLR
51 и 142
0,037
0,604
INHBC_LDFHFSSDR_vs_TENX_LSQLSVTDVTTSSLR
107 и 142
0,009
0,630
BGH3_LTLLAPLNSVFK_vs_PRG2_WNFAYWAAHQPWSR
52 и 129
0,035
0,605
INHBC_LDFHFSSDR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
107 и 144
0,024
0,612
BGH3_LTLLAPLNSVFK_vs_PSG3_VSAPSGTGHLPGLNPL
52 и 134
0,043
0,601
INHBC_LDFHFSSDR_vs_VTDB_ELPEHTVK
107 и 147
0,025
0,612
C1QB_VPGLYYFTYHASSR_vs_IGF2_GIVEECCFR
55 и 103
0,022
0,614
ITIH3_ALDLSLK_vs_PRG2_WNFAYWAAHQPWSR
111 и 129
0,024
0,613
C1QB_VPGLYYFTYHASSR_vs_PRG2_WNFAYWAAHQPWSR
55 и 129
0,011
0,627
ITIH3_ALDLSLK_vs_PSG3_VSAPSGTGHLPGLNPL
111 и 134
0,042
0,602
C1QB_VPGLYYFTYHASSR_vs_PSG3_VSAPSGTGHLPGLNPL
55 и 134
0,008
0,633
ITIH3_ALDLSLK_vs_SHBG_IALGGLLFPASNLR
111 и 18
0,024
0,612
C1QB_VPGLYYFTYHASSR_vs_SHBG_IALGGLLFPASNLR
55 и 18
0,012
0,625
ITIH4_NPLVWVHASPEHVVVTR_vs_PRG2_WNFAYWAAHQPWSR
113 и 129
0,020
0,616
C1QB_VPGLYYFTYHASSR_vs_TENX_LNWEAPPGAFDSFLLR
55 и 141
0,031
0,608
ITIH4_NPLVWVHASPEHVVVTR_vs_PSG3_VSAPSGTGHLPGLNPL
113 и 134
0,016
0,620
C1QB_VPGLYYFTYHASSR_vs_TENX_LSQLSVTDVTTSSLR
55 и 142
0,040
0,602
ITIH4_NPLVWVHASPEHVVVTR_vs_SHBG_IALGGLLFPASNLR
113 и 18
0,034
0,606
CATD_VGFAEAAR_vs_PRG2_WNFAYWAAHQPWSR
57 и 129
0,033
0,607
KNG1_DIPTNSPELEETLTHTITK_vs_IGF2_GIVEECCFR
116 и 103
0,030
0,609
CD14_LTVGAAQVPAQLLVGALR_vs_IGF2_GIVEECCFR
61 и 103
0,011
0,626
KNG1_DIPTNSPELEETLTHTITK_vs_PRG2_WNFAYWAAHQPWSR
116 и 129
0,032
0,607
CD14_LTVGAAQVPAQLLVGALR_vs_PRG2_WNFAYWAAHQPWSR
61 и 129
0,028
0,610
KNG1_DIPTNSPELEETLTHTITK_vs_PSG3_VSAPSGTGHLPGLNPL
116 и 134
0,020
0,617
CD14_LTVGAAQVPAQLLVGALR_vs_PSG3_VSAPSGTGHLPGLNPL
61 и 134
0,008
0,633
KNG1_DIPTNSPELEETLTHTITK_vs_SHBG_IALGGLLFPASNLR
116 и 18
0,041
0,602
CD14_LTVGAAQVPAQLLVGALR_vs_SHBG_IALGGLLFPASNLR
61 и 18
0,022
0,615
KNG1_DIPTNSPELEETLTHTITK_vs_TENX_LSQLSVTDVTTSSLR
116 и 142
0,043
0,601
CD14_LTVGAAQVPAQLLVGALR_vs_TENX_LNWEAPPGAFDSFLLR
61 и 141
0,027
0,611
KNG1_QVVAGLNFR_vs_IGF2_GIVEECCFR
117 и 103
0,032
0,607
CD14_LTVGAAQVPAQLLVGALR_vs_TENX_LSQLSVTDVTTSSLR
61 и 142
0,028
0,610
KNG1_QVVAGLNFR_vs_PRG2_WNFAYWAAHQPWSR
117 и 129
0,035
0,606
CD14_SWLAELQQWLKPGLK_vs_IGF2_GIVEECCFR
62 и 103
0,034
0,606
KNG1_QVVAGLNFR_vs_PSG3_VSAPSGTGHLPGLNPL
117 и 134
0,014
0,623
CD14_SWLAELQQWLKPGLK_vs_PRG2_WNFAYWAAHQPWSR
62 и 129
0,033
0,607
KNG1_QVVAGLNFR_vs_SHBG_IALGGLLFPASNLR
117 и 18
0,025
0,612
CD14_SWLAELQQWLKPGLK_vs_PSG3_VSAPSGTGHLPGLNPL
62 и 134
0,014
0,622
LBP_ITGFLKPGK_vs_CRIS3_YEDLYSNCK
118 и 79
0,044
0,600
CD14_SWLAELQQWLKPGLK_vs_SHBG_IALGGLLFPASNLR
62 и 18
0,034
0,606
LBP_ITGFLKPGK_vs_IGF2_GIVEECCFR
118 и 103
0,043
0,601
CD14_SWLAELQQWLKPGLK_vs_TENX_LNWEAPPGAFDSFLLR
62 и 141
0,042
0,602
LBP_ITGFLKPGK_vs_PRG2_WNFAYWAAHQPWSR
118 и 129
0,022
0,615
CFAB_YGLVTYATYPK_vs_IGF2_GIVEECCFR
64 и 103
0,030
0,609
LBP_ITGFLKPGK_vs_PSG3_VSAPSGTGHLPGLNPL
118 и 134
0,006
0,637
CFAB_YGLVTYATYPK_vs_PRG2_WNFAYWAAHQPWSR
64 и 129
0,037
0,604
LBP_ITGFLKPGK_vs_SHBG_IALGGLLFPASNLR
118 и 18
0,033
0,606
CFAB_YGLVTYATYPK_vs_PSG3_VSAPSGTGHLPGLNPL
64 и 134
0,022
0,615
LBP_ITLPDFTGDLR_vs_CRIS3_AVSPPAR
119 и 78
0,024
0,613
CLUS_ASSIIDELFQDR_vs_PSG3_VSAPSGTGHLPGLNPL
67 и 134
0,036
0,605
LBP_ITLPDFTGDLR_vs_CRIS3_YEDLYSNCK
119 и 79
0,018
0,618
CO5_TLLPVSKPEIR_vs_PRG2_WNFAYWAAHQPWSR
70 и 129
0,037
0,604
LBP_ITLPDFTGDLR_vs_IGF2_GIVEECCFR
119 и 103
0,024
0,613
CO5_TLLPVSKPEIR_vs_PSG3_VSAPSGTGHLPGLNPL
70 и 134
0,017
0,619
LBP_ITLPDFTGDLR_vs_PRG2_WNFAYWAAHQPWSR
119 и 129
0,011
0,628
CO5_VFQFLEK_vs_PRG2_WNFAYWAAHQPWSR
71 и 129
0,031
0,608
LBP_ITLPDFTGDLR_vs_PSG3_VSAPSGTGHLPGLNPL
119 и 134
0,002
0,652
CO5_VFQFLEK_vs_PSG3_VSAPSGTGHLPGLNPL
71 и 134
0,023
0,614
LBP_ITLPDFTGDLR_vs_SHBG_IALGGLLFPASNLR
119 и 18
0,011
0,628
CO5_VFQFLEK_vs_SHBG_IALGGLLFPASNLR
71 и 18
0,035
0,605
PEDF_LQSLFDSPDFSK_vs_PSG3_VSAPSGTGHLPGLNPL
124 и 134
0,015
0,621
CO6_ALNHLPLEYNSALYSR_vs_PSG3_VSAPSGTGHLPGLNPL
72 и 134
0,034
0,606
PEDF_LQSLFDSPDFSK_vs_TENX_LSQLSVTDVTTSSLR
124 и 142
0,045
0,600
CO8A_SLLQPNK_vs_IBP3_FLNVLSPR
74 и 99
0,044
0,600
PEDF_TVQAVLTVPK_vs_PRG2_WNFAYWAAHQPWSR
125 и 129
0,037
0,604
CO8A_SLLQPNK_vs_IBP3_YGQPLPGYTTK
74 и 100
0,026
0,611
PEDF_TVQAVLTVPK_vs_PSG3_VSAPSGTGHLPGLNPL
125 и 134
0,017
0,619
CO8A_SLLQPNK_vs_IGF2_GIVEECCFR
74 и 103
0,004
0,643
PRDX2_GLFIIDGK_vs_PRG2_WNFAYWAAHQPWSR
128 и 129
0,018
0,618
CO8A_SLLQPNK_vs_PRG2_WNFAYWAAHQPWSR
74 и 129
0,021
0,615
PRDX2_GLFIIDGK_vs_PSG3_VSAPSGTGHLPGLNPL
128 и 134
0,019
0,617
CO8A_SLLQPNK_vs_PSG3_VSAPSGTGHLPGLNPL
74 и 134
0,009
0,631
PRDX2_GLFIIDGK_vs_SHBG_IALGGLLFPASNLR
128 и 18
0,026
0,611
CO8A_SLLQPNK_vs_SHBG_IALGGLLFPASNLR
74 и 18
0,019
0,617
PRDX2_GLFIIDGK_vs_TENX_LNWEAPPGAFDSFLLR
128 и 141
0,022
0,614
CO8A_SLLQPNK_vs_SPRL1_VLTHSELAPLR
74 и 140
0,035
0,605
PRDX2_GLFIIDGK_vs_TENX_LSQLSVTDVTTSSLR
128 и 142
0,042
0,602
CO8A_SLLQPNK_vs_TENX_LNWEAPPGAFDSFLLR
74 и 141
0,024
0,613
PTGDS_GPGEDFR_vs_PRG2_WNFAYWAAHQPWSR
137 и 129
0,026
0,611
CO8A_SLLQPNK_vs_TENX_LSQLSVTDVTTSSLR
74 и 142
0,019
0,617
PTGDS_GPGEDFR_vs_PSG3_VSAPSGTGHLPGLNPL
137 и 134
0,028
0,610
CO8A_SLLQPNK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
74 и 144
0,026
0,611
THBG_AVLHIGEK_vs_PRG2_WNFAYWAAHQPWSR
143 и 129
0,029
0,609
CO8B_QALEEFQK_vs_CRIS3_YEDLYSNCK
76 и 79
0,038
0,604
THBG_AVLHIGEK_vs_PSG3_VSAPSGTGHLPGLNPL
143 и 134
0,018
0,618
CO8B_QALEEFQK_vs_IBP3_FLNVLSPR
76 и 99
0,025
0,612
THBG_AVLHIGEK_vs_SHBG_IALGGLLFPASNLR
143 и 18
0,033
0,606
CO8B_QALEEFQK_vs_IBP3_YGQPLPGYTTK
76 и 100
0,019
0,618
VTNC_GQYCYELDEK_vs_IGF2_GIVEECCFR
149 и 103
0,030
0,609
CO8B_QALEEFQK_vs_IGF2_GIVEECCFR
76 и 103
0,002
0,654
VTNC_GQYCYELDEK_vs_PRG2_WNFAYWAAHQPWSR
149 и 129
0,018
0,618
CO8B_QALEEFQK_vs_PRG2_WNFAYWAAHQPWSR
76 и 129
0,021
0,616
VTNC_GQYCYELDEK_vs_PSG3_VSAPSGTGHLPGLNPL
149 и 134
0,014
0,622
CO8B_QALEEFQK_vs_PSG3_VSAPSGTGHLPGLNPL
76 и 134
0,004
0,645
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,009
0,631
CO8B_QALEEFQK_vs_SHBG_IALGGLLFPASNLR
76 и 18
0,020
0,616
VTNC_VDTVDPPYPR_vs_IBP3_YGQPLPGYTTK
150 и 100
0,033
0,607
CO8B_QALEEFQK_vs_SPRL1_VLTHSELAPLR
76 и 140
0,021
0,615
VTNC_VDTVDPPYPR_vs_IGF2_GIVEECCFR
150 и 103
0,013
0,624
CO8B_QALEEFQK_vs_TENX_LNWEAPPGAFDSFLLR
76 и 141
0,015
0,622
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,033
0,606
CO8B_QALEEFQK_vs_TENX_LSQLSVTDVTTSSLR
76 и 142
0,010
0,628
VTNC_VDTVDPPYPR_vs_PRG2_WNFAYWAAHQPWSR
150 и 129
0,014
0,623
CO8B_QALEEFQK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
76 и 144
0,017
0,620
VTNC_VDTVDPPYPR_vs_PSG3_VSAPSGTGHLPGLNPL
150 и 134
0,004
0,642
CO8B_QALEEFQK_vs_VTDB_ELPEHTVK
76 и 147
0,041
0,602
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,004
0,643
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PRG2_WNFAYWAAHQPWSR
82 и 129
0,033
0,607
VTNC_VDTVDPPYPR_vs_TENX_LNWEAPPGAFDSFLLR
150 и 141
0,032
0,607
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_SHBG_IALGGLLFPASNLR
82 и 18
0,033
0,606
VTNC_VDTVDPPYPR_vs_TENX_LSQLSVTDVTTSSLR
150 и 142
0,043
0,601
ENPP2_TYLHTYESEI_vs_PRG2_WNFAYWAAHQPWSR
83 и 129
0,040
0,603
VTNC_VDTVDPPYPR_vs_VTDB_ELPEHTVK
150 и 147
0,015
0,621
ENPP2_TYLHTYESEI_vs_PSG3_VSAPSGTGHLPGLNPL
83 и 134
0,044
0,601
Таблица 36: эффективность классификации по обращениям, недели 19 и 20.
AUROC обращения для недель беременности 19 и 20 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, с BMI-стратификацией (>22≤37).
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
A2GL_DLLLPQPDLR_vs_CRIS3_AVSPPAR
34 и 78
0,041
0,628
CO8B_QALEEFQK_vs_TENX_LNWEAPPGAFDSFLLR
76 и 141
0,008
0,665
A2GL_DLLLPQPDLR_vs_CRIS3_YEDLYSNCK
34 и 79
0,024
0,641
CO8B_QALEEFQK_vs_TENX_LSQLSVTDVTTSSLR
76 и 142
0,005
0,674
A2GL_DLLLPQPDLR_vs_LYAM1_SYYWIGIR
34 и 120
0,034
0,633
CO8B_QALEEFQK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
76 и 144
0,024
0,642
A2GL_DLLLPQPDLR_vs_PSG3_VSAPSGTGHLPGLNPL
34 и 134
0,011
0,659
CO8B_QALEEFQK_vs_VTDB_ELPEHTVK
76 и 147
0,020
0,645
A2GL_DLLLPQPDLR_vs_SHBG_IALGGLLFPASNLR
34 и 18
0,030
0,636
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_SHBG_IALGGLLFPASNLR
82 и 18
0,046
0,625
AFAM_DADPDTFFAK_vs_IBP3_YGQPLPGYTTK
37 и 100
0,044
0,626
F13B_GDTYPAELYITGSILR_vs_CRIS3_YEDLYSNCK
84 и 79
0,040
0,628
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,036
0,631
FETUA_FSVVYAK_vs_PSG3_VSAPSGTGHLPGLNPL
88 и 134
0,038
0,630
AFAM_DADPDTFFAK_vs_PSG3_VSAPSGTGHLPGLNPL
37 и 134
0,028
0,637
FETUA_HTLNQIDEVK_vs_PSG3_VSAPSGTGHLPGLNPL
89 и 134
0,040
0,629
AFAM_HFQNLGK_vs_IBP3_FLNVLSPR
38 и 99
0,040
0,628
IBP4_QCHPALDGQR_vs_CRIS3_AVSPPAR
2 и 78
0,002
0,693
AFAM_HFQNLGK_vs_IBP3_YGQPLPGYTTK
38 и 100
0,018
0,647
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,001
0,704
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,018
0,648
IBP4_QCHPALDGQR_vs_IBP3_FLNVLSPR
2 и 99
0,034
0,632
AFAM_HFQNLGK_vs_PSG3_VSAPSGTGHLPGLNPL
38 и 134
0,014
0,654
IBP4_QCHPALDGQR_vs_IBP3_YGQPLPGYTTK
2 и 100
0,010
0,660
AFAM_HFQNLGK_vs_TENX_LNWEAPPGAFDSFLLR
38 и 141
0,047
0,624
IBP4_QCHPALDGQR_vs_IGF2_GIVEECCFR
2 и 103
0,003
0,687
ANGT_DPTFIPAPIQAK_vs_CRIS3_YEDLYSNCK
42 и 79
0,047
0,624
IBP4_QCHPALDGQR_vs_ITIH4_ILDDLSPR
2 и 112
0,014
0,654
ANGT_DPTFIPAPIQAK_vs_PSG3_VSAPSGTGHLPGLNPL
42 и 134
0,025
0,640
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,002
0,695
APOC3_GWVTDGFSSLK_vs_C163A_INPASLDK
47 и 54
0,035
0,632
IBP4_QCHPALDGQR_vs_NCAM1_GLGEISAASEFK
2 и 121
0,023
0,642
APOC3_GWVTDGFSSLK_vs_CRIS3_AVSPPAR
47 и 78
0,011
0,658
IBP4_QCHPALDGQR_vs_PGRP2_AGLLRPDYALLGHR
2 и 126
0,016
0,651
APOC3_GWVTDGFSSLK_vs_CRIS3_YEDLYSNCK
47 и 79
0,009
0,663
IBP4_QCHPALDGQR_vs_PSG3_VSAPSGTGHLPGLNPL
2 и 134
0,000
0,741
APOC3_GWVTDGFSSLK_vs_IBP3_FLNVLSPR
47 и 99
0,032
0,634
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,002
0,690
APOC3_GWVTDGFSSLK_vs_IBP3_YGQPLPGYTTK
47 и 100
0,035
0,632
IBP4_QCHPALDGQR_vs_SPRL1_VLTHSELAPLR
2 и 140
0,006
0,671
APOC3_GWVTDGFSSLK_vs_IGF2_GIVEECCFR
47 и 103
0,024
0,642
IBP4_QCHPALDGQR_vs_TENX_LNWEAPPGAFDSFLLR
2 и 141
0,005
0,674
APOC3_GWVTDGFSSLK_vs_ITIH4_ILDDLSPR
47 и 112
0,046
0,625
IBP4_QCHPALDGQR_vs_TENX_LSQLSVTDVTTSSLR
2 и 142
0,009
0,662
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,015
0,652
IBP4_QCHPALDGQR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
2 и 144
0,012
0,657
APOC3_GWVTDGFSSLK_vs_PSG3_VSAPSGTGHLPGLNPL
47 и 134
0,010
0,661
IBP4_QCHPALDGQR_vs_VTDB_ELPEHTVK
2 и 147
0,003
0,686
APOC3_GWVTDGFSSLK_vs_SHBG_IALGGLLFPASNLR
47 и 18
0,024
0,641
INHBC_LDFHFSSDR_vs_ALS_IRPHTFTGLSGLR
107 и 40
0,024
0,642
APOC3_GWVTDGFSSLK_vs_SOM2.CSH_SVEGSCGF
47 и 139
0,049
0,623
INHBC_LDFHFSSDR_vs_CRIS3_AVSPPAR
107 и 78
0,029
0,637
APOC3_GWVTDGFSSLK_vs_SPRL1_VLTHSELAPLR
47 и 140
0,049
0,623
INHBC_LDFHFSSDR_vs_CRIS3_YEDLYSNCK
107 и 79
0,015
0,652
APOC3_GWVTDGFSSLK_vs_TENX_LNWEAPPGAFDSFLLR
47 и 141
0,048
0,624
INHBC_LDFHFSSDR_vs_IBP3_FLNVLSPR
107 и 99
0,007
0,669
APOC3_GWVTDGFSSLK_vs_TENX_LSQLSVTDVTTSSLR
47 и 142
0,049
0,623
INHBC_LDFHFSSDR_vs_IBP3_YGQPLPGYTTK
107 и 100
0,005
0,674
APOC3_GWVTDGFSSLK_vs_VTDB_ELPEHTVK
47 и 147
0,042
0,627
INHBC_LDFHFSSDR_vs_IGF2_GIVEECCFR
107 и 103
0,002
0,695
APOH_ATVVYQGER_vs_CRIS3_YEDLYSNCK
48 и 79
0,037
0,631
INHBC_LDFHFSSDR_vs_ITIH4_ILDDLSPR
107 и 112
0,033
0,633
APOH_ATVVYQGER_vs_IBP3_YGQPLPGYTTK
48 и 100
0,039
0,629
INHBC_LDFHFSSDR_vs_LYAM1_SYYWIGIR
107 и 120
0,030
0,636
APOH_ATVVYQGER_vs_IGF2_GIVEECCFR
48 и 103
0,046
0,625
INHBC_LDFHFSSDR_vs_NCAM1_GLGEISAASEFK
107 и 121
0,047
0,624
APOH_ATVVYQGER_vs_PSG3_VSAPSGTGHLPGLNPL
48 и 134
0,013
0,656
INHBC_LDFHFSSDR_vs_PGRP2_AGLLRPDYALLGHR
107 и 126
0,036
0,631
B2MG_VNHVTLSQPK_vs_CRIS3_YEDLYSNCK
51 и 79
0,025
0,640
INHBC_LDFHFSSDR_vs_PSG3_VSAPSGTGHLPGLNPL
107 и 134
0,001
0,710
B2MG_VNHVTLSQPK_vs_IGF2_GIVEECCFR
51 и 103
0,038
0,630
INHBC_LDFHFSSDR_vs_SHBG_IALGGLLFPASNLR
107 и 18
0,008
0,666
B2MG_VNHVTLSQPK_vs_PSG3_VSAPSGTGHLPGLNPL
51 и 134
0,028
0,637
INHBC_LDFHFSSDR_vs_SPRL1_VLTHSELAPLR
107 и 140
0,019
0,646
BGH3_LTLLAPLNSVFK_vs_CRIS3_YEDLYSNCK
52 и 79
0,036
0,631
INHBC_LDFHFSSDR_vs_TENX_LNWEAPPGAFDSFLLR
107 и 141
0,002
0,693
BGH3_LTLLAPLNSVFK_vs_PSG3_VSAPSGTGHLPGLNPL
52 и 134
0,031
0,635
INHBC_LDFHFSSDR_vs_TENX_LSQLSVTDVTTSSLR
107 и 142
0,003
0,689
C1QB_VPGLYYFTYHASSR_vs_PSG3_VSAPSGTGHLPGLNPL
55 и 134
0,019
0,647
INHBC_LDFHFSSDR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
107 и 144
0,029
0,637
C1QB_VPGLYYFTYHASSR_vs_SHBG_IALGGLLFPASNLR
55 и 18
0,047
0,624
INHBC_LDFHFSSDR_vs_VTDB_ELPEHTVK
107 и 147
0,008
0,666
CAH1_GGPFSDSYR_vs_CRIS3_YEDLYSNCK
56 и 79
0,036
0,631
ITIH3_ALDLSLK_vs_CRIS3_YEDLYSNCK
111 и 79
0,044
0,626
CAH1_GGPFSDSYR_vs_PSG3_VSAPSGTGHLPGLNPL
56 и 134
0,028
0,637
KNG1_DIPTNSPELEETLTHTITK_vs_CRIS3_YEDLYSNCK
116 и 79
0,040
0,628
CBPN_NNANGVDLNR_vs_PSG3_VSAPSGTGHLPGLNPL
60 и 134
0,023
0,643
KNG1_QVVAGLNFR_vs_CRIS3_YEDLYSNCK
117 и 79
0,049
0,623
CD14_LTVGAAQVPAQLLVGALR_vs_CRIS3_YEDLYSNCK
61 и 79
0,034
0,633
KNG1_QVVAGLNFR_vs_PSG3_VSAPSGTGHLPGLNPL
117 и 134
0,033
0,633
CD14_LTVGAAQVPAQLLVGALR_vs_PSG3_VSAPSGTGHLPGLNPL
61 и 134
0,029
0,637
LBP_ITGFLKPGK_vs_PSG3_VSAPSGTGHLPGLNPL
118 и 134
0,021
0,644
CD14_SWLAELQQWLKPGLK_vs_PSG3_VSAPSGTGHLPGLNPL
62 и 134
0,040
0,628
LBP_ITLPDFTGDLR_vs_CRIS3_AVSPPAR
119 и 78
0,031
0,635
CFAB_YGLVTYATYPK_vs_PSG3_VSAPSGTGHLPGLNPL
64 и 134
0,020
0,646
LBP_ITLPDFTGDLR_vs_CRIS3_YEDLYSNCK
119 и 79
0,021
0,645
CLUS_ASSIIDELFQDR_vs_PSG3_VSAPSGTGHLPGLNPL
67 и 134
0,027
0,638
LBP_ITLPDFTGDLR_vs_PSG3_VSAPSGTGHLPGLNPL
119 и 134
0,008
0,666
CO5_TLLPVSKPEIR_vs_CRIS3_AVSPPAR
70 и 78
0,049
0,623
LBP_ITLPDFTGDLR_vs_SHBG_IALGGLLFPASNLR
119 и 18
0,021
0,644
CO5_TLLPVSKPEIR_vs_CRIS3_YEDLYSNCK
70 и 79
0,039
0,629
PEDF_LQSLFDSPDFSK_vs_PSG3_VSAPSGTGHLPGLNPL
124 и 134
0,036
0,631
CO5_TLLPVSKPEIR_vs_PSG3_VSAPSGTGHLPGLNPL
70 и 134
0,010
0,661
PEDF_TVQAVLTVPK_vs_PSG3_VSAPSGTGHLPGLNPL
125 и 134
0,017
0,649
CO5_VFQFLEK_vs_CRIS3_YEDLYSNCK
71 и 79
0,041
0,628
PRDX2_GLFIIDGK_vs_CRIS3_AVSPPAR
128 и 78
0,019
0,647
CO5_VFQFLEK_vs_PSG3_VSAPSGTGHLPGLNPL
71 и 134
0,028
0,637
PRDX2_GLFIIDGK_vs_CRIS3_YEDLYSNCK
128 и 79
0,012
0,658
CO6_ALNHLPLEYNSALYSR_vs_PSG3_VSAPSGTGHLPGLNPL
72 и 134
0,050
0,623
PRDX2_GLFIIDGK_vs_LYAM1_SYYWIGIR
128 и 120
0,028
0,637
CO8A_SLLQPNK_vs_CRIS3_AVSPPAR
74 и 78
0,039
0,629
PRDX2_GLFIIDGK_vs_PGRP2_AGLLRPDYALLGHR
128 и 126
0,049
0,623
CO8A_SLLQPNK_vs_CRIS3_YEDLYSNCK
74 и 79
0,016
0,651
PRDX2_GLFIIDGK_vs_PSG3_VSAPSGTGHLPGLNPL
128 и 134
0,005
0,676
CO8A_SLLQPNK_vs_IBP3_YGQPLPGYTTK
74 и 100
0,038
0,630
PRDX2_GLFIIDGK_vs_SHBG_IALGGLLFPASNLR
128 и 18
0,021
0,645
CO8A_SLLQPNK_vs_IGF2_GIVEECCFR
74 и 103
0,015
0,652
PRDX2_GLFIIDGK_vs_SPRL1_VLTHSELAPLR
128 и 140
0,049
0,623
CO8A_SLLQPNK_vs_PSG3_VSAPSGTGHLPGLNPL
74 и 134
0,004
0,680
PRDX2_GLFIIDGK_vs_TENX_LNWEAPPGAFDSFLLR
128 и 141
0,017
0,650
CO8A_SLLQPNK_vs_SHBG_IALGGLLFPASNLR
74 и 18
0,031
0,635
PRDX2_GLFIIDGK_vs_TENX_LSQLSVTDVTTSSLR
128 и 142
0,030
0,636
CO8A_SLLQPNK_vs_SPRL1_VLTHSELAPLR
74 и 140
0,018
0,648
PSG2_IHPSYTNYR_vs_PSG3_VSAPSGTGHLPGLNPL
133 и 134
0,044
0,626
CO8A_SLLQPNK_vs_TENX_LNWEAPPGAFDSFLLR
74 и 141
0,022
0,644
PTGDS_GPGEDFR_vs_CRIS3_YEDLYSNCK
137 и 79
0,037
0,630
CO8A_SLLQPNK_vs_TENX_LSQLSVTDVTTSSLR
74 и 142
0,017
0,650
PTGDS_GPGEDFR_vs_IGF2_GIVEECCFR
137 и 103
0,048
0,624
CO8B_QALEEFQK_vs_ALS_IRPHTFTGLSGLR
76 и 40
0,035
0,632
PTGDS_GPGEDFR_vs_PSG3_VSAPSGTGHLPGLNPL
137 и 134
0,020
0,645
CO8B_QALEEFQK_vs_CRIS3_AVSPPAR
76 и 78
0,017
0,649
THBG_AVLHIGEK_vs_PSG3_VSAPSGTGHLPGLNPL
143 и 134
0,026
0,639
CO8B_QALEEFQK_vs_CRIS3_YEDLYSNCK
76 и 79
0,008
0,665
VTNC_GQYCYELDEK_vs_CRIS3_YEDLYSNCK
149 и 79
0,037
0,630
CO8B_QALEEFQK_vs_IBP3_FLNVLSPR
76 и 99
0,028
0,638
VTNC_GQYCYELDEK_vs_PSG3_VSAPSGTGHLPGLNPL
149 и 134
0,013
0,655
CO8B_QALEEFQK_vs_IBP3_YGQPLPGYTTK
76 и 100
0,017
0,649
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,024
0,641
CO8B_QALEEFQK_vs_IGF2_GIVEECCFR
76 и 103
0,005
0,677
VTNC_VDTVDPPYPR_vs_CRIS3_AVSPPAR
150 и 78
0,032
0,634
CO8B_QALEEFQK_vs_LYAM1_SYYWIGIR
76 и 120
0,039
0,629
VTNC_VDTVDPPYPR_vs_CRIS3_YEDLYSNCK
150 и 79
0,023
0,643
CO8B_QALEEFQK_vs_NCAM1_GLGEISAASEFK
76 и 121
0,035
0,632
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,036
0,631
CO8B_QALEEFQK_vs_PSG3_VSAPSGTGHLPGLNPL
76 и 134
0,002
0,698
VTNC_VDTVDPPYPR_vs_PSG3_VSAPSGTGHLPGLNPL
150 и 134
0,005
0,675
CO8B_QALEEFQK_vs_SHBG_IALGGLLFPASNLR
76 и 18
0,027
0,639
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,013
0,656
CO8B_QALEEFQK_vs_SPRL1_VLTHSELAPLR
76 и 140
0,008
0,666
Таблица 37: эффективность классификации по обращениям, недели 19 и 20.
AUROC обращения для недель беременности 19 и 20 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, без BMI-стратификации.
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
AFAM_DADPDTFFAK_vs_LYAM1_SYYWIGIR
37 и 120
0,047
0,659
CD14_SWLAELQQWLKPGLK_vs_PGRP2_AGLLRPDYALLGHR
62 и 126
0,014
0,697
AFAM_DADPDTFFAK_vs_SHBG_IALGGLLFPASNLR
37 и 18
0,043
0,663
CD14_SWLAELQQWLKPGLK_vs_PSG3_VSAPSGTGHLPGLNPL
62 и 134
0,031
0,673
AFAM_HFQNLGK_vs_LYAM1_SYYWIGIR
38 и 120
0,040
0,664
CD14_SWLAELQQWLKPGLK_vs_SHBG_IALGGLLFPASNLR
62 и 18
0,011
0,703
APOC3_GWVTDGFSSLK_vs_ALS_IRPHTFTGLSGLR
47 и 40
0,018
0,690
CD14_SWLAELQQWLKPGLK_vs_SPRL1_VLTHSELAPLR
62 и 140
0,021
0,685
APOC3_GWVTDGFSSLK_vs_C163A_INPASLDK
47 и 54
0,024
0,680
CD14_SWLAELQQWLKPGLK_vs_TENX_LNWEAPPGAFDSFLLR
62 и 141
0,042
0,663
APOC3_GWVTDGFSSLK_vs_CRIS3_AVSPPAR
47 и 78
0,008
0,714
CD14_SWLAELQQWLKPGLK_vs_TENX_LSQLSVTDVTTSSLR
62 и 142
0,028
0,676
APOC3_GWVTDGFSSLK_vs_CRIS3_YEDLYSNCK
47 и 79
0,009
0,710
CD14_SWLAELQQWLKPGLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
62 и 144
0,026
0,679
APOC3_GWVTDGFSSLK_vs_CSH_AHQLAIDTYQEFEETYIPK
47 и 80
0,018
0,689
CD14_SWLAELQQWLKPGLK_vs_VTDB_ELPEHTVK
62 и 147
0,007
0,718
APOC3_GWVTDGFSSLK_vs_CSH_ISLLLIESWLEPVR
47 и 81
0,025
0,680
CLUS_ASSIIDELFQDR_vs_FBLN1_TGYYFDGISR
67 и 86
0,048
0,658
APOC3_GWVTDGFSSLK_vs_FBLN1_TGYYFDGISR
47 и 86
0,017
0,691
CLUS_ASSIIDELFQDR_vs_LYAM1_SYYWIGIR
67 и 120
0,004
0,728
APOC3_GWVTDGFSSLK_vs_IBP1_VVESLAK
47 и 97
0,022
0,683
CLUS_ASSIIDELFQDR_vs_PGRP2_AGLLRPDYALLGHR
67 и 126
0,036
0,668
APOC3_GWVTDGFSSLK_vs_IBP3_FLNVLSPR
47 и 99
0,019
0,688
CLUS_ASSIIDELFQDR_vs_SHBG_IALGGLLFPASNLR
67 и 18
0,008
0,713
APOC3_GWVTDGFSSLK_vs_IBP3_YGQPLPGYTTK
47 и 100
0,020
0,686
CLUS_ASSIIDELFQDR_vs_SPRL1_VLTHSELAPLR
67 и 140
0,031
0,673
APOC3_GWVTDGFSSLK_vs_IGF2_GIVEECCFR
47 и 103
0,026
0,679
CLUS_ASSIIDELFQDR_vs_TENX_LSQLSVTDVTTSSLR
67 и 142
0,045
0,660
APOC3_GWVTDGFSSLK_vs_ITIH4_ILDDLSPR
47 и 112
0,016
0,694
CLUS_ASSIIDELFQDR_vs_VTDB_ELPEHTVK
67 и 147
0,015
0,695
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,001
0,756
CLUS_LFDSDPITVTVPVEVSR_vs_CRIS3_AVSPPAR
68 и 78
0,034
0,670
APOC3_GWVTDGFSSLK_vs_NCAM1_GLGEISAASEFK
47 и 121
0,031
0,673
CLUS_LFDSDPITVTVPVEVSR_vs_CRIS3_YEDLYSNCK
68 и 79
0,028
0,676
APOC3_GWVTDGFSSLK_vs_PGRP2_AGLLRPDYALLGHR
47 и 126
0,009
0,711
CLUS_LFDSDPITVTVPVEVSR_vs_FBLN1_TGYYFDGISR
68 и 86
0,036
0,668
APOC3_GWVTDGFSSLK_vs_PSG1_FQLPGQK
47 и 131
0,027
0,677
CLUS_LFDSDPITVTVPVEVSR_vs_LYAM1_SYYWIGIR
68 и 120
0,001
0,767
APOC3_GWVTDGFSSLK_vs_PSG3_VSAPSGTGHLPGLNPL
47 и 134
0,021
0,684
CLUS_LFDSDPITVTVPVEVSR_vs_PGRP2_AGLLRPDYALLGHR
68 и 126
0,013
0,699
APOC3_GWVTDGFSSLK_vs_SHBG_IALGGLLFPASNLR
47 и 18
0,008
0,713
CLUS_LFDSDPITVTVPVEVSR_vs_PSG3_VSAPSGTGHLPGLNPL
68 и 134
0,020
0,686
APOC3_GWVTDGFSSLK_vs_SOM2.CSH_NYGLLYCFR
47 и 138
0,029
0,675
CLUS_LFDSDPITVTVPVEVSR_vs_SHBG_IALGGLLFPASNLR
68 и 18
0,004
0,730
APOC3_GWVTDGFSSLK_vs_SOM2.CSH_SVEGSCGF
47 и 139
0,020
0,687
CLUS_LFDSDPITVTVPVEVSR_vs_SPRL1_VLTHSELAPLR
68 и 140
0,012
0,702
APOC3_GWVTDGFSSLK_vs_SPRL1_VLTHSELAPLR
47 и 140
0,014
0,697
CLUS_LFDSDPITVTVPVEVSR_vs_TENX_LNWEAPPGAFDSFLLR
68 и 141
0,021
0,684
APOC3_GWVTDGFSSLK_vs_TENX_LNWEAPPGAFDSFLLR
47 и 141
0,017
0,691
CLUS_LFDSDPITVTVPVEVSR_vs_TENX_LSQLSVTDVTTSSLR
68 и 142
0,013
0,698
APOC3_GWVTDGFSSLK_vs_TENX_LSQLSVTDVTTSSLR
47 и 142
0,015
0,694
CLUS_LFDSDPITVTVPVEVSR_vs_VTDB_ELPEHTVK
68 и 147
0,002
0,753
APOC3_GWVTDGFSSLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
47 и 144
0,012
0,702
CO5_TLLPVSKPEIR_vs_LYAM1_SYYWIGIR
70 и 120
0,009
0,710
APOC3_GWVTDGFSSLK_vs_VTDB_ELPEHTVK
47 и 147
0,010
0,708
CO5_TLLPVSKPEIR_vs_SHBG_IALGGLLFPASNLR
70 и 18
0,027
0,677
APOH_ATVVYQGER_vs_ALS_IRPHTFTGLSGLR
48 и 40
0,039
0,666
CO5_TLLPVSKPEIR_vs_SPRL1_VLTHSELAPLR
70 и 140
0,028
0,676
APOH_ATVVYQGER_vs_CRIS3_AVSPPAR
48 и 78
0,018
0,690
CO5_VFQFLEK_vs_LYAM1_SYYWIGIR
71 и 120
0,013
0,699
APOH_ATVVYQGER_vs_CRIS3_YEDLYSNCK
48 и 79
0,013
0,698
CO5_VFQFLEK_vs_SHBG_IALGGLLFPASNLR
71 и 18
0,030
0,674
APOH_ATVVYQGER_vs_FBLN1_TGYYFDGISR
48 и 86
0,029
0,675
CO6_ALNHLPLEYNSALYSR_vs_LYAM1_SYYWIGIR
72 и 120
0,017
0,692
APOH_ATVVYQGER_vs_IBP1_VVESLAK
48 и 97
0,033
0,671
CO6_ALNHLPLEYNSALYSR_vs_SHBG_IALGGLLFPASNLR
72 и 18
0,040
0,665
APOH_ATVVYQGER_vs_IBP3_YGQPLPGYTTK
48 и 100
0,040
0,664
CO8A_SLLQPNK_vs_LYAM1_SYYWIGIR
74 и 120
0,009
0,711
APOH_ATVVYQGER_vs_IGF2_GIVEECCFR
48 и 103
0,045
0,660
CO8A_SLLQPNK_vs_PGRP2_AGLLRPDYALLGHR
74 и 126
0,045
0,661
APOH_ATVVYQGER_vs_ITIH4_ILDDLSPR
48 и 112
0,020
0,686
CO8A_SLLQPNK_vs_SHBG_IALGGLLFPASNLR
74 и 18
0,033
0,671
APOH_ATVVYQGER_vs_LYAM1_SYYWIGIR
48 и 120
0,001
0,762
CO8A_SLLQPNK_vs_SPRL1_VLTHSELAPLR
74 и 140
0,036
0,668
APOH_ATVVYQGER_vs_NCAM1_GLGEISAASEFK
48 и 121
0,047
0,659
CO8A_SLLQPNK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
74 и 144
0,040
0,665
APOH_ATVVYQGER_vs_PGRP2_AGLLRPDYALLGHR
48 и 126
0,003
0,736
CO8B_QALEEFQK_vs_LYAM1_SYYWIGIR
76 и 120
0,009
0,711
APOH_ATVVYQGER_vs_PSG3_VSAPSGTGHLPGLNPL
48 и 134
0,005
0,725
CO8B_QALEEFQK_vs_PSG3_VSAPSGTGHLPGLNPL
76 и 134
0,035
0,669
APOH_ATVVYQGER_vs_SHBG_IALGGLLFPASNLR
48 и 18
0,005
0,726
CO8B_QALEEFQK_vs_SHBG_IALGGLLFPASNLR
76 и 18
0,036
0,668
APOH_ATVVYQGER_vs_SPRL1_VLTHSELAPLR
48 и 140
0,007
0,718
CO8B_QALEEFQK_vs_SPRL1_VLTHSELAPLR
76 и 140
0,025
0,679
APOH_ATVVYQGER_vs_TENX_LNWEAPPGAFDSFLLR
48 и 141
0,010
0,705
CO8B_QALEEFQK_vs_TENX_LSQLSVTDVTTSSLR
76 и 142
0,043
0,662
APOH_ATVVYQGER_vs_TENX_LSQLSVTDVTTSSLR
48 и 142
0,012
0,702
CO8B_QALEEFQK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
76 и 144
0,032
0,672
APOH_ATVVYQGER_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
48 и 144
0,041
0,664
ENPP2_TYLHTYESEI_vs_LYAM1_SYYWIGIR
83 и 120
0,043
0,662
APOH_ATVVYQGER_vs_VTDB_ELPEHTVK
48 и 147
0,002
0,749
F13B_GDTYPAELYITGSILR_vs_CRIS3_AVSPPAR
84 и 78
0,024
0,681
B2MG_VEHSDLSFSK_vs_LYAM1_SYYWIGIR
50 и 120
0,018
0,690
F13B_GDTYPAELYITGSILR_vs_CRIS3_YEDLYSNCK
84 и 79
0,022
0,683
B2MG_VNHVTLSQPK_vs_CRIS3_AVSPPAR
51 и 78
0,047
0,659
F13B_GDTYPAELYITGSILR_vs_FBLN1_TGYYFDGISR
84 и 86
0,046
0,660
B2MG_VNHVTLSQPK_vs_CRIS3_YEDLYSNCK
51 и 79
0,047
0,659
F13B_GDTYPAELYITGSILR_vs_LYAM1_SYYWIGIR
84 и 120
0,001
0,754
B2MG_VNHVTLSQPK_vs_LYAM1_SYYWIGIR
51 и 120
0,003
0,735
F13B_GDTYPAELYITGSILR_vs_PGRP2_AGLLRPDYALLGHR
84 и 126
0,037
0,667
B2MG_VNHVTLSQPK_vs_PGRP2_AGLLRPDYALLGHR
51 и 126
0,039
0,666
F13B_GDTYPAELYITGSILR_vs_SHBG_IALGGLLFPASNLR
84 и 18
0,011
0,703
B2MG_VNHVTLSQPK_vs_SHBG_IALGGLLFPASNLR
51 и 18
0,019
0,688
F13B_GDTYPAELYITGSILR_vs_SPRL1_VLTHSELAPLR
84 и 140
0,016
0,694
B2MG_VNHVTLSQPK_vs_SPRL1_VLTHSELAPLR
51 и 140
0,037
0,667
F13B_GDTYPAELYITGSILR_vs_TENX_LNWEAPPGAFDSFLLR
84 и 141
0,046
0,660
BGH3_LTLLAPLNSVFK_vs_LYAM1_SYYWIGIR
52 и 120
0,025
0,679
F13B_GDTYPAELYITGSILR_vs_TENX_LSQLSVTDVTTSSLR
84 и 142
0,030
0,673
CAH1_GGPFSDSYR_vs_CRIS3_AVSPPAR
56 и 78
0,026
0,679
F13B_GDTYPAELYITGSILR_vs_VTDB_ELPEHTVK
84 и 147
0,043
0,662
CAH1_GGPFSDSYR_vs_CRIS3_YEDLYSNCK
56 и 79
0,029
0,675
HABP2_FLNWIK_vs_LYAM1_SYYWIGIR
92 и 120
0,017
0,692
CAH1_GGPFSDSYR_vs_IBP1_VVESLAK
56 и 97
0,036
0,668
HABP2_FLNWIK_vs_PGRP2_AGLLRPDYALLGHR
92 и 126
0,027
0,677
CAH1_GGPFSDSYR_vs_LYAM1_SYYWIGIR
56 и 120
0,015
0,694
HABP2_FLNWIK_vs_SHBG_IALGGLLFPASNLR
92 и 18
0,020
0,687
CAH1_GGPFSDSYR_vs_PGRP2_AGLLRPDYALLGHR
56 и 126
0,032
0,672
HABP2_FLNWIK_vs_TENX_LNWEAPPGAFDSFLLR
92 и 141
0,033
0,671
CAH1_GGPFSDSYR_vs_SHBG_IALGGLLFPASNLR
56 и 18
0,017
0,691
HABP2_FLNWIK_vs_TENX_LSQLSVTDVTTSSLR
92 и 142
0,030
0,673
CAH1_GGPFSDSYR_vs_TENX_LNWEAPPGAFDSFLLR
56 и 141
0,028
0,676
HABP2_FLNWIK_vs_VTDB_ELPEHTVK
92 и 147
0,022
0,684
CAH1_GGPFSDSYR_vs_TENX_LSQLSVTDVTTSSLR
56 и 142
0,039
0,665
HEMO_NFPSPVDAAFR_vs_LYAM1_SYYWIGIR
93 и 120
0,020
0,686
CATD_VGFAEAAR_vs_C163A_INPASLDK
57 и 54
0,006
0,719
HEMO_NFPSPVDAAFR_vs_SHBG_IALGGLLFPASNLR
93 и 18
0,022
0,684
CATD_VGFAEAAR_vs_CRIS3_AVSPPAR
57 и 78
0,012
0,700
IBP4_QCHPALDGQR_vs_CRIS3_AVSPPAR
2 и 78
0,006
0,720
CATD_VGFAEAAR_vs_CRIS3_YEDLYSNCK
57 и 79
0,014
0,696
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,009
0,708
CATD_VGFAEAAR_vs_CSH_AHQLAIDTYQEFEETYIPK
57 и 80
0,011
0,704
IBP4_QCHPALDGQR_vs_FBLN1_TGYYFDGISR
2 и 86
0,040
0,664
CATD_VGFAEAAR_vs_CSH_ISLLLIESWLEPVR
57 и 81
0,016
0,692
IBP4_QCHPALDGQR_vs_IBP1_VVESLAK
2 и 97
0,038
0,666
CATD_VGFAEAAR_vs_FBLN1_TGYYFDGISR
57 и 86
0,012
0,701
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,001
0,761
CATD_VGFAEAAR_vs_IBP1_VVESLAK
57 и 97
0,048
0,659
IBP4_QCHPALDGQR_vs_PGRP2_AGLLRPDYALLGHR
2 и 126
0,026
0,678
CATD_VGFAEAAR_vs_IGF2_GIVEECCFR
57 и 103
0,034
0,670
IBP4_QCHPALDGQR_vs_PSG3_VSAPSGTGHLPGLNPL
2 и 134
0,013
0,698
CATD_VGFAEAAR_vs_ITIH4_ILDDLSPR
57 и 112
0,026
0,679
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,005
0,725
CATD_VGFAEAAR_vs_LYAM1_SYYWIGIR
57 и 120
0,001
0,758
IBP4_QCHPALDGQR_vs_SPRL1_VLTHSELAPLR
2 и 140
0,007
0,715
CATD_VGFAEAAR_vs_NCAM1_GLGEISAASEFK
57 и 121
0,035
0,669
IBP4_QCHPALDGQR_vs_TENX_LNWEAPPGAFDSFLLR
2 и 141
0,047
0,659
CATD_VGFAEAAR_vs_PGRP2_AGLLRPDYALLGHR
57 и 126
0,006
0,720
IBP4_QCHPALDGQR_vs_TENX_LSQLSVTDVTTSSLR
2 и 142
0,044
0,661
CATD_VGFAEAAR_vs_PRG2_WNFAYWAAHQPWSR
57 и 129
0,048
0,658
IBP4_QCHPALDGQR_vs_VTDB_ELPEHTVK
2 и 147
0,033
0,671
CATD_VGFAEAAR_vs_PSG1_FQLPGQK
57 и 131
0,008
0,713
IBP6_GAQTLYVPNCDHR_vs_LYAM1_SYYWIGIR
101 и 120
0,049
0,657
CATD_VGFAEAAR_vs_PSG3_VSAPSGTGHLPGLNPL
57 и 134
0,015
0,695
IBP6_GAQTLYVPNCDHR_vs_SHBG_IALGGLLFPASNLR
101 и 18
0,033
0,671
CATD_VGFAEAAR_vs_SHBG_IALGGLLFPASNLR
57 и 18
0,006
0,719
IBP6_HLDSVLQQLQTEVYR_vs_LYAM1_SYYWIGIR
102 и 120
0,026
0,679
CATD_VGFAEAAR_vs_SOM2.CSH_NYGLLYCFR
57 и 138
0,028
0,676
IBP6_HLDSVLQQLQTEVYR_vs_SHBG_IALGGLLFPASNLR
102 и 18
0,033
0,671
CATD_VGFAEAAR_vs_SOM2.CSH_SVEGSCGF
57 и 139
0,020
0,687
INHBC_LDFHFSSDR_vs_LYAM1_SYYWIGIR
107 и 120
0,038
0,666
CATD_VGFAEAAR_vs_SPRL1_VLTHSELAPLR
57 и 140
0,007
0,716
INHBC_LDFHFSSDR_vs_PGRP2_AGLLRPDYALLGHR
107 и 126
0,044
0,661
CATD_VGFAEAAR_vs_TENX_LNWEAPPGAFDSFLLR
57 и 141
0,004
0,730
INHBC_LDFHFSSDR_vs_SHBG_IALGGLLFPASNLR
107 и 18
0,043
0,662
CATD_VGFAEAAR_vs_TENX_LSQLSVTDVTTSSLR
57 и 142
0,004
0,733
INHBC_LDFHFSSDR_vs_TENX_LNWEAPPGAFDSFLLR
107 и 141
0,047
0,659
CATD_VGFAEAAR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
57 и 144
0,016
0,693
INHBC_LDFHFSSDR_vs_TENX_LSQLSVTDVTTSSLR
107 и 142
0,047
0,659
CATD_VGFAEAAR_vs_VTDB_ELPEHTVK
57 и 147
0,019
0,687
KNG1_DIPTNSPELEETLTHTITK_vs_LYAM1_SYYWIGIR
116 и 120
0,013
0,700
CATD_VSTLPAITLK_vs_ALS_IRPHTFTGLSGLR
58 и 40
0,031
0,673
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,003
0,734
CATD_VSTLPAITLK_vs_C163A_INPASLDK
58 и 54
0,026
0,678
KNG1_QVVAGLNFR_vs_PGRP2_AGLLRPDYALLGHR
117 и 126
0,048
0,659
CATD_VSTLPAITLK_vs_CRIS3_AVSPPAR
58 и 78
0,012
0,702
KNG1_QVVAGLNFR_vs_SHBG_IALGGLLFPASNLR
117 и 18
0,012
0,702
CATD_VSTLPAITLK_vs_CRIS3_YEDLYSNCK
58 и 79
0,014
0,696
KNG1_QVVAGLNFR_vs_SPRL1_VLTHSELAPLR
117 и 140
0,020
0,687
CATD_VSTLPAITLK_vs_CSH_AHQLAIDTYQEFEETYIPK
58 и 80
0,022
0,683
PEDF_LQSLFDSPDFSK_vs_CRIS3_AVSPPAR
124 и 78
0,022
0,683
CATD_VSTLPAITLK_vs_CSH_ISLLLIESWLEPVR
58 и 81
0,034
0,670
PEDF_LQSLFDSPDFSK_vs_CRIS3_YEDLYSNCK
124 и 79
0,026
0,679
CATD_VSTLPAITLK_vs_FBLN1_TGYYFDGISR
58 и 86
0,011
0,704
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,005
0,726
CATD_VSTLPAITLK_vs_IBP3_FLNVLSPR
58 и 99
0,033
0,671
PEDF_LQSLFDSPDFSK_vs_SHBG_IALGGLLFPASNLR
124 и 18
0,045
0,660
CATD_VSTLPAITLK_vs_IBP3_YGQPLPGYTTK
58 и 100
0,029
0,675
PEDF_TVQAVLTVPK_vs_CRIS3_AVSPPAR
125 и 78
0,023
0,683
CATD_VSTLPAITLK_vs_IGF2_GIVEECCFR
58 и 103
0,014
0,697
PEDF_TVQAVLTVPK_vs_CRIS3_YEDLYSNCK
125 и 79
0,024
0,681
CATD_VSTLPAITLK_vs_ITIH4_ILDDLSPR
58 и 112
0,030
0,674
PEDF_TVQAVLTVPK_vs_LYAM1_SYYWIGIR
125 и 120
0,003
0,736
CATD_VSTLPAITLK_vs_LYAM1_SYYWIGIR
58 и 120
0,002
0,749
PEDF_TVQAVLTVPK_vs_PSG3_VSAPSGTGHLPGLNPL
125 и 134
0,047
0,659
CATD_VSTLPAITLK_vs_NCAM1_GLGEISAASEFK
58 и 121
0,024
0,681
PEDF_TVQAVLTVPK_vs_SHBG_IALGGLLFPASNLR
125 и 18
0,024
0,681
CATD_VSTLPAITLK_vs_PGRP2_AGLLRPDYALLGHR
58 и 126
0,005
0,727
PEDF_TVQAVLTVPK_vs_SPRL1_VLTHSELAPLR
125 и 140
0,029
0,675
CATD_VSTLPAITLK_vs_PSG1_FQLPGQK
58 и 131
0,025
0,679
PEDF_TVQAVLTVPK_vs_TENX_LSQLSVTDVTTSSLR
125 и 142
0,038
0,667
CATD_VSTLPAITLK_vs_PSG3_VSAPSGTGHLPGLNPL
58 и 134
0,009
0,710
PEDF_TVQAVLTVPK_vs_VTDB_ELPEHTVK
125 и 147
0,036
0,668
CATD_VSTLPAITLK_vs_SHBG_IALGGLLFPASNLR
58 и 18
0,005
0,723
PRDX2_GLFIIDGK_vs_CRIS3_AVSPPAR
128 и 78
0,032
0,672
CATD_VSTLPAITLK_vs_SOM2.CSH_NYGLLYCFR
58 и 138
0,039
0,665
PRDX2_GLFIIDGK_vs_CRIS3_YEDLYSNCK
128 и 79
0,029
0,675
CATD_VSTLPAITLK_vs_SOM2.CSH_SVEGSCGF
58 и 139
0,046
0,660
PRDX2_GLFIIDGK_vs_IBP1_VVESLAK
128 и 97
0,033
0,671
CATD_VSTLPAITLK_vs_SPRL1_VLTHSELAPLR
58 и 140
0,006
0,720
PRDX2_GLFIIDGK_vs_LYAM1_SYYWIGIR
128 и 120
0,013
0,700
CATD_VSTLPAITLK_vs_TENX_LNWEAPPGAFDSFLLR
58 и 141
0,003
0,741
PRDX2_GLFIIDGK_vs_PGRP2_AGLLRPDYALLGHR
128 и 126
0,028
0,676
CATD_VSTLPAITLK_vs_TENX_LSQLSVTDVTTSSLR
58 и 142
0,003
0,737
PRDX2_GLFIIDGK_vs_PSG3_VSAPSGTGHLPGLNPL
128 и 134
0,047
0,659
CATD_VSTLPAITLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
58 и 144
0,026
0,679
PRDX2_GLFIIDGK_vs_SHBG_IALGGLLFPASNLR
128 и 18
0,012
0,702
CATD_VSTLPAITLK_vs_VTDB_ELPEHTVK
58 и 147
0,007
0,717
PRDX2_GLFIIDGK_vs_TENX_LNWEAPPGAFDSFLLR
128 и 141
0,020
0,687
CBPN_EALIQFLEQVHQGIK_vs_SHBG_IALGGLLFPASNLR
59 и 18
0,038
0,667
PRDX2_GLFIIDGK_vs_TENX_LSQLSVTDVTTSSLR
128 и 142
0,028
0,676
CBPN_NNANGVDLNR_vs_LYAM1_SYYWIGIR
60 и 120
0,032
0,672
PSG2_IHPSYTNYR_vs_FBLN1_TGYYFDGISR
133 и 86
0,023
0,682
CBPN_NNANGVDLNR_vs_SPRL1_VLTHSELAPLR
60 и 140
0,018
0,690
PSG2_IHPSYTNYR_vs_LYAM1_SYYWIGIR
133 и 120
0,043
0,662
CD14_LTVGAAQVPAQLLVGALR_vs_CRIS3_AVSPPAR
61 и 78
0,042
0,663
PSG2_IHPSYTNYR_vs_SPRL1_VLTHSELAPLR
133 и 140
0,049
0,657
CD14_LTVGAAQVPAQLLVGALR_vs_CRIS3_YEDLYSNCK
61 и 79
0,036
0,668
PTGDS_GPGEDFR_vs_CRIS3_AVSPPAR
137 и 78
0,031
0,673
CD14_LTVGAAQVPAQLLVGALR_vs_CSH_AHQLAIDTYQEFEETYIPK
61 и 80
0,043
0,663
PTGDS_GPGEDFR_vs_CRIS3_YEDLYSNCK
137 и 79
0,035
0,669
CD14_LTVGAAQVPAQLLVGALR_vs_FBLN1_TGYYFDGISR
61 и 86
0,022
0,684
PTGDS_GPGEDFR_vs_FBLN1_TGYYFDGISR
137 и 86
0,032
0,672
CD14_LTVGAAQVPAQLLVGALR_vs_IBP1_VVESLAK
61 и 97
0,044
0,661
PTGDS_GPGEDFR_vs_LYAM1_SYYWIGIR
137 и 120
0,007
0,715
CD14_LTVGAAQVPAQLLVGALR_vs_LYAM1_SYYWIGIR
61 и 120
0,001
0,759
PTGDS_GPGEDFR_vs_PGRP2_AGLLRPDYALLGHR
137 и 126
0,044
0,661
CD14_LTVGAAQVPAQLLVGALR_vs_NCAM1_GLGEISAASEFK
61 и 121
0,043
0,663
PTGDS_GPGEDFR_vs_SHBG_IALGGLLFPASNLR
137 и 18
0,033
0,671
CD14_LTVGAAQVPAQLLVGALR_vs_PGRP2_AGLLRPDYALLGHR
61 и 126
0,011
0,704
PTGDS_GPGEDFR_vs_SOM2.CSH_SVEGSCGF
137 и 139
0,041
0,664
CD14_LTVGAAQVPAQLLVGALR_vs_PSG3_VSAPSGTGHLPGLNPL
61 и 134
0,020
0,687
PTGDS_GPGEDFR_vs_SPRL1_VLTHSELAPLR
137 и 140
0,016
0,692
CD14_LTVGAAQVPAQLLVGALR_vs_SHBG_IALGGLLFPASNLR
61 и 18
0,008
0,713
THBG_AVLHIGEK_vs_LYAM1_SYYWIGIR
143 и 120
0,013
0,698
CD14_LTVGAAQVPAQLLVGALR_vs_SPRL1_VLTHSELAPLR
61 и 140
0,015
0,695
THBG_AVLHIGEK_vs_SHBG_IALGGLLFPASNLR
143 и 18
0,041
0,664
CD14_LTVGAAQVPAQLLVGALR_vs_TENX_LNWEAPPGAFDSFLLR
61 и 141
0,026
0,678
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,015
0,696
CD14_LTVGAAQVPAQLLVGALR_vs_TENX_LSQLSVTDVTTSSLR
61 и 142
0,019
0,688
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,031
0,672
CD14_LTVGAAQVPAQLLVGALR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
61 и 144
0,022
0,683
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,008
0,712
CD14_LTVGAAQVPAQLLVGALR_vs_VTDB_ELPEHTVK
61 и 147
0,005
0,727
VTNC_VDTVDPPYPR_vs_PGRP2_AGLLRPDYALLGHR
150 и 126
0,040
0,664
CD14_SWLAELQQWLKPGLK_vs_FBLN1_TGYYFDGISR
62 и 86
0,033
0,671
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,014
0,696
CD14_SWLAELQQWLKPGLK_vs_LYAM1_SYYWIGIR
62 и 120
0,002
0,743
VTNC_VDTVDPPYPR_vs_VTDB_ELPEHTVK
150 и 147
0,030
0,673
Таблица 38: эффективность классификации по обращениям, недели 19 и 20.
AUROC обращения для недель беременности 19 и 20 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, с BMI-стратификацией (>22≤37).
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
AFAM_DADPDTFFAK_vs_CRIS3_YEDLYSNCK
37 и 79
0,048
0,709
CLUS_LFDSDPITVTVPVEVSR_vs_SHBG_IALGGLLFPASNLR
68 и 18
0,009
0,776
AFAM_DADPDTFFAK_vs_LYAM1_SYYWIGIR
37 и 120
0,027
0,734
CLUS_LFDSDPITVTVPVEVSR_vs_VTDB_ELPEHTVK
68 и 147
0,035
0,723
AFAM_DADPDTFFAK_vs_PGRP2_AGLLRPDYALLGHR
37 и 126
0,007
0,788
CO5_TLLPVSKPEIR_vs_LYAM1_SYYWIGIR
70 и 120
0,033
0,726
AFAM_DADPDTFFAK_vs_SHBG_IALGGLLFPASNLR
37 и 18
0,010
0,771
CO5_TLLPVSKPEIR_vs_SHBG_IALGGLLFPASNLR
70 и 18
0,018
0,750
AFAM_DADPDTFFAK_vs_VTDB_ELPEHTVK
37 и 147
0,027
0,734
CO5_VFQFLEK_vs_SHBG_IALGGLLFPASNLR
71 и 18
0,031
0,728
AFAM_HFQNLGK_vs_IBP1_VVESLAK
38 и 97
0,045
0,726
CO6_ALNHLPLEYNSALYSR_vs_LYAM1_SYYWIGIR
72 и 120
0,041
0,716
AFAM_HFQNLGK_vs_LYAM1_SYYWIGIR
38 и 120
0,021
0,744
CO6_ALNHLPLEYNSALYSR_vs_SHBG_IALGGLLFPASNLR
72 и 18
0,024
0,739
AFAM_HFQNLGK_vs_PGRP2_AGLLRPDYALLGHR
38 и 126
0,012
0,766
CO8A_SLLQPNK_vs_LYAM1_SYYWIGIR
74 и 120
0,029
0,731
AFAM_HFQNLGK_vs_SHBG_IALGGLLFPASNLR
38 и 18
0,016
0,756
CO8A_SLLQPNK_vs_PGRP2_AGLLRPDYALLGHR
74 и 126
0,044
0,713
AFAM_HFQNLGK_vs_VTDB_ELPEHTVK
38 и 147
0,036
0,722
CO8A_SLLQPNK_vs_SHBG_IALGGLLFPASNLR
74 и 18
0,034
0,725
ANGT_DPTFIPAPIQAK_vs_SHBG_IALGGLLFPASNLR
42 и 18
0,025
0,737
CO8B_QALEEFQK_vs_LYAM1_SYYWIGIR
76 и 120
0,028
0,733
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,033
0,726
CO8B_QALEEFQK_vs_SHBG_IALGGLLFPASNLR
76 и 18
0,041
0,716
APOH_ATVVYQGER_vs_CRIS3_AVSPPAR
48 и 78
0,020
0,746
F13B_GDTYPAELYITGSILR_vs_CRIS3_AVSPPAR
84 и 78
0,015
0,757
APOH_ATVVYQGER_vs_CRIS3_YEDLYSNCK
48 и 79
0,013
0,762
F13B_GDTYPAELYITGSILR_vs_CRIS3_YEDLYSNCK
84 и 79
0,016
0,756
APOH_ATVVYQGER_vs_ITIH4_ILDDLSPR
48 и 112
0,043
0,714
F13B_GDTYPAELYITGSILR_vs_LYAM1_SYYWIGIR
84 и 120
0,003
0,815
APOH_ATVVYQGER_vs_LYAM1_SYYWIGIR
48 и 120
0,003
0,816
F13B_GDTYPAELYITGSILR_vs_PGRP2_AGLLRPDYALLGHR
84 и 126
0,015
0,758
APOH_ATVVYQGER_vs_PGRP2_AGLLRPDYALLGHR
48 и 126
0,001
0,855
F13B_GDTYPAELYITGSILR_vs_SHBG_IALGGLLFPASNLR
84 и 18
0,004
0,801
APOH_ATVVYQGER_vs_SHBG_IALGGLLFPASNLR
48 и 18
0,003
0,819
F13B_GDTYPAELYITGSILR_vs_VTDB_ELPEHTVK
84 и 147
0,023
0,740
APOH_ATVVYQGER_vs_SPRL1_VLTHSELAPLR
48 и 140
0,043
0,714
FBLN3_IPSNPSHR_vs_CHL1_VIAVNEVGR
87 и 66
0,036
0,722
APOH_ATVVYQGER_vs_VTDB_ELPEHTVK
48 и 147
0,001
0,839
HEMO_NFPSPVDAAFR_vs_SHBG_IALGGLLFPASNLR
93 и 18
0,036
0,722
B2MG_VNHVTLSQPK_vs_LYAM1_SYYWIGIR
51 и 120
0,008
0,782
IBP4_QCHPALDGQR_vs_CRIS3_AVSPPAR
2 и 78
0,008
0,780
B2MG_VNHVTLSQPK_vs_SHBG_IALGGLLFPASNLR
51 и 18
0,022
0,742
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,012
0,766
BGH3_LTLLAPLNSVFK_vs_LYAM1_SYYWIGIR
52 и 120
0,028
0,732
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,001
0,861
BGH3_LTLLAPLNSVFK_vs_PGRP2_AGLLRPDYALLGHR
52 и 126
0,034
0,725
IBP4_QCHPALDGQR_vs_PGRP2_AGLLRPDYALLGHR
2 и 126
0,014
0,761
BGH3_LTLLAPLNSVFK_vs_SHBG_IALGGLLFPASNLR
52 и 18
0,027
0,734
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,002
0,827
BGH3_LTLLAPLNSVFK_vs_SOM2.CSH_SVEGSCGF
52 и 139
0,040
0,717
IBP4_QCHPALDGQR_vs_VTDB_ELPEHTVK
2 и 147
0,021
0,745
CAH1_GGPFSDSYR_vs_CRIS3_AVSPPAR
56 и 78
0,031
0,729
IBP6_GAQTLYVPNCDHR_vs_LYAM1_SYYWIGIR
101 и 120
0,050
0,708
CAH1_GGPFSDSYR_vs_CRIS3_YEDLYSNCK
56 и 79
0,042
0,715
IBP6_HLDSVLQQLQTEVYR_vs_LYAM1_SYYWIGIR
102 и 120
0,013
0,763
CAH1_GGPFSDSYR_vs_LYAM1_SYYWIGIR
56 и 120
0,033
0,726
IBP6_HLDSVLQQLQTEVYR_vs_SHBG_IALGGLLFPASNLR
102 и 18
0,026
0,735
CAH1_GGPFSDSYR_vs_PGRP2_AGLLRPDYALLGHR
56 и 126
0,045
0,712
INHBC_LDFHFSSDR_vs_PGRP2_AGLLRPDYALLGHR
107 и 126
0,043
0,714
CAH1_GGPFSDSYR_vs_SHBG_IALGGLLFPASNLR
56 и 18
0,020
0,747
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,013
0,762
CATD_VGFAEAAR_vs_C163A_INPASLDK
57 и 54
0,035
0,723
KNG1_QVVAGLNFR_vs_SHBG_IALGGLLFPASNLR
117 и 18
0,033
0,726
CATD_VGFAEAAR_vs_CRIS3_AVSPPAR
57 и 78
0,029
0,731
PAPP1_DIPHWLNPTR_vs_CRIS3_AVSPPAR
122 и 78
0,048
0,709
CATD_VGFAEAAR_vs_CRIS3_YEDLYSNCK
57 и 79
0,038
0,720
PAPP1_DIPHWLNPTR_vs_CRIS3_YEDLYSNCK
122 и 79
0,045
0,712
CATD_VGFAEAAR_vs_CSH_AHQLAIDTYQEFEETYIPK
57 и 80
0,028
0,733
PAPP1_DIPHWLNPTR_vs_CSH_ISLLLIESWLEPVR
122 и 81
0,048
0,709
CATD_VGFAEAAR_vs_CSH_ISLLLIESWLEPVR
57 и 81
0,038
0,720
PAPP1_DIPHWLNPTR_vs_LYAM1_SYYWIGIR
122 и 120
0,021
0,745
CATD_VGFAEAAR_vs_LYAM1_SYYWIGIR
57 и 120
0,006
0,789
PAPP1_DIPHWLNPTR_vs_PRG2_WNFAYWAAHQPWSR
122 и 129
0,033
0,726
CATD_VGFAEAAR_vs_PGRP2_AGLLRPDYALLGHR
57 и 126
0,018
0,751
PAPP1_DIPHWLNPTR_vs_PSG1_FQLPGQK
122 и 131
0,040
0,718
CATD_VGFAEAAR_vs_SHBG_IALGGLLFPASNLR
57 и 18
0,013
0,762
PAPP1_DIPHWLNPTR_vs_SHBG_IALGGLLFPASNLR
122 и 18
0,035
0,723
CATD_VGFAEAAR_vs_SOM2.CSH_SVEGSCGF
57 и 139
0,044
0,713
PAPP1_DIPHWLNPTR_vs_SOM2.CSH_SVEGSCGF
122 и 139
0,018
0,750
CATD_VGFAEAAR_vs_TENX_LNWEAPPGAFDSFLLR
57 и 141
0,045
0,712
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,015
0,758
CATD_VSTLPAITLK_vs_CRIS3_AVSPPAR
58 и 78
0,016
0,754
PEDF_LQSLFDSPDFSK_vs_SHBG_IALGGLLFPASNLR
124 и 18
0,024
0,739
CATD_VSTLPAITLK_vs_CRIS3_YEDLYSNCK
58 и 79
0,014
0,760
PEDF_LQSLFDSPDFSK_vs_VTDB_ELPEHTVK
124 и 147
0,048
0,709
CATD_VSTLPAITLK_vs_CSH_AHQLAIDTYQEFEETYIPK
58 и 80
0,029
0,731
PEDF_TVQAVLTVPK_vs_CRIS3_AVSPPAR
125 и 78
0,047
0,710
CATD_VSTLPAITLK_vs_CSH_ISLLLIESWLEPVR
58 и 81
0,041
0,716
PEDF_TVQAVLTVPK_vs_LYAM1_SYYWIGIR
125 и 120
0,008
0,783
CATD_VSTLPAITLK_vs_ITIH4_ILDDLSPR
58 и 112
0,024
0,739
PEDF_TVQAVLTVPK_vs_SHBG_IALGGLLFPASNLR
125 и 18
0,009
0,777
CATD_VSTLPAITLK_vs_LYAM1_SYYWIGIR
58 и 120
0,002
0,822
PEDF_TVQAVLTVPK_vs_VTDB_ELPEHTVK
125 и 147
0,031
0,729
CATD_VSTLPAITLK_vs_NCAM1_GLGEISAASEFK
58 и 121
0,045
0,712
PRDX2_GLFIIDGK_vs_SHBG_IALGGLLFPASNLR
128 и 18
0,019
0,748
CATD_VSTLPAITLK_vs_PGRP2_AGLLRPDYALLGHR
58 и 126
0,004
0,809
PSG2_IHPSYTNYR_vs_CRIS3_AVSPPAR
133 и 78
0,016
0,756
CATD_VSTLPAITLK_vs_SHBG_IALGGLLFPASNLR
58 и 18
0,005
0,795
PSG2_IHPSYTNYR_vs_CRIS3_YEDLYSNCK
133 и 79
0,018
0,750
CATD_VSTLPAITLK_vs_SPRL1_VLTHSELAPLR
58 и 140
0,037
0,721
PSG2_IHPSYTNYR_vs_IBP1_VVESLAK
133 и 97
0,030
0,730
CATD_VSTLPAITLK_vs_TENX_LNWEAPPGAFDSFLLR
58 и 141
0,014
0,759
PSG2_IHPSYTNYR_vs_IBP2_LIQGAPTIR
133 и 98
0,026
0,736
CATD_VSTLPAITLK_vs_TENX_LSQLSVTDVTTSSLR
58 и 142
0,021
0,745
PSG2_IHPSYTNYR_vs_ITIH4_ILDDLSPR
133 и 112
0,019
0,749
CATD_VSTLPAITLK_vs_VTDB_ELPEHTVK
58 и 147
0,013
0,764
PSG2_IHPSYTNYR_vs_LYAM1_SYYWIGIR
133 и 120
0,007
0,788
CBPN_NNANGVDLNR_vs_SHBG_IALGGLLFPASNLR
60 и 18
0,040
0,718
PSG2_IHPSYTNYR_vs_PGRP2_AGLLRPDYALLGHR
133 и 126
0,022
0,742
CD14_LTVGAAQVPAQLLVGALR_vs_CRIS3_AVSPPAR
61 и 78
0,041
0,716
PSG2_IHPSYTNYR_vs_PSG3_VSAPSGTGHLPGLNPL
133 и 134
0,024
0,739
CD14_LTVGAAQVPAQLLVGALR_vs_CRIS3_YEDLYSNCK
61 и 79
0,028
0,732
PSG2_IHPSYTNYR_vs_SHBG_IALGGLLFPASNLR
133 и 18
0,005
0,794
CD14_LTVGAAQVPAQLLVGALR_vs_LYAM1_SYYWIGIR
61 и 120
0,005
0,799
PSG2_IHPSYTNYR_vs_SOM2.CSH_SVEGSCGF
133 и 139
0,036
0,722
CD14_LTVGAAQVPAQLLVGALR_vs_PGRP2_AGLLRPDYALLGHR
61 и 126
0,012
0,766
PSG2_IHPSYTNYR_vs_SPRL1_VLTHSELAPLR
133 и 140
0,022
0,743
CD14_LTVGAAQVPAQLLVGALR_vs_SHBG_IALGGLLFPASNLR
61 и 18
0,011
0,768
PSG2_IHPSYTNYR_vs_TENX_LNWEAPPGAFDSFLLR
133 и 141
0,028
0,733
CD14_LTVGAAQVPAQLLVGALR_vs_VTDB_ELPEHTVK
61 и 147
0,016
0,756
PSG2_IHPSYTNYR_vs_TENX_LSQLSVTDVTTSSLR
133 и 142
0,029
0,731
CD14_SWLAELQQWLKPGLK_vs_LYAM1_SYYWIGIR
62 и 120
0,015
0,758
PSG2_IHPSYTNYR_vs_VTDB_ELPEHTVK
133 и 147
0,013
0,764
CD14_SWLAELQQWLKPGLK_vs_PGRP2_AGLLRPDYALLGHR
62 и 126
0,024
0,739
PTGDS_GPGEDFR_vs_CRIS3_AVSPPAR
137 и 78
0,020
0,747
CD14_SWLAELQQWLKPGLK_vs_SHBG_IALGGLLFPASNLR
62 и 18
0,017
0,752
PTGDS_GPGEDFR_vs_CRIS3_YEDLYSNCK
137 и 79
0,019
0,749
CD14_SWLAELQQWLKPGLK_vs_VTDB_ELPEHTVK
62 и 147
0,034
0,725
PTGDS_GPGEDFR_vs_LYAM1_SYYWIGIR
137 и 120
0,003
0,814
CLUS_ASSIIDELFQDR_vs_CRIS3_AVSPPAR
67 и 78
0,035
0,723
PTGDS_GPGEDFR_vs_PGRP2_AGLLRPDYALLGHR
137 и 126
0,012
0,766
CLUS_ASSIIDELFQDR_vs_CRIS3_YEDLYSNCK
67 и 79
0,040
0,718
PTGDS_GPGEDFR_vs_SHBG_IALGGLLFPASNLR
137 и 18
0,019
0,748
CLUS_ASSIIDELFQDR_vs_LYAM1_SYYWIGIR
67 и 120
0,018
0,751
PTGDS_GPGEDFR_vs_SOM2.CSH_SVEGSCGF
137 и 139
0,010
0,774
CLUS_ASSIIDELFQDR_vs_PGRP2_AGLLRPDYALLGHR
67 и 126
0,042
0,715
PTGDS_GPGEDFR_vs_VTDB_ELPEHTVK
137 и 147
0,027
0,734
CLUS_ASSIIDELFQDR_vs_SHBG_IALGGLLFPASNLR
67 и 18
0,013
0,763
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,024
0,739
CLUS_ASSIIDELFQDR_vs_VTDB_ELPEHTVK
67 и 147
0,040
0,717
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,030
0,730
CLUS_LFDSDPITVTVPVEVSR_vs_CRIS3_AVSPPAR
68 и 78
0,031
0,729
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,025
0,738
CLUS_LFDSDPITVTVPVEVSR_vs_CRIS3_YEDLYSNCK
68 и 79
0,031
0,728
VTNC_VDTVDPPYPR_vs_PGRP2_AGLLRPDYALLGHR
150 и 126
0,046
0,711
CLUS_LFDSDPITVTVPVEVSR_vs_LYAM1_SYYWIGIR
68 и 120
0,011
0,768
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,012
0,765
CLUS_LFDSDPITVTVPVEVSR_vs_PGRP2_AGLLRPDYALLGHR
68 и 126
0,031
0,728
68 и 18
Таблица 39: эффективность классификации по обращениям, недели 20 и 21.
AUROC обращения для недель беременности 20 и 21 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, без BMI-стратификации.
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
A2GL_DLLLPQPDLR_vs_IGF2_GIVEECCFR
34 и 103
0,028
0,636
ENPP2_TYLHTYESEI_vs_LYAM1_SYYWIGIR
83 и 120
0,001
0,712
A2GL_DLLLPQPDLR_vs_SHBG_IALGGLLFPASNLR
34 и 18
0,035
0,631
ENPP2_TYLHTYESEI_vs_NCAM1_GLGEISAASEFK
83 и 121
0,010
0,660
A2GL_DLLLPQPDLR_vs_TENX_LNWEAPPGAFDSFLLR
34 и 141
0,035
0,631
ENPP2_TYLHTYESEI_vs_PGRP2_AGLLRPDYALLGHR
83 и 126
0,001
0,714
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,011
0,659
ENPP2_TYLHTYESEI_vs_PRG2_WNFAYWAAHQPWSR
83 и 129
0,003
0,686
AFAM_DADPDTFFAK_vs_TENX_LNWEAPPGAFDSFLLR
37 и 141
0,027
0,638
ENPP2_TYLHTYESEI_vs_PSG1_FQLPGQK
83 и 131
0,047
0,623
AFAM_HFQNLGK_vs_CHL1_VIAVNEVGR
38 и 66
0,036
0,631
ENPP2_TYLHTYESEI_vs_PSG3_VSAPSGTGHLPGLNPL
83 и 134
0,004
0,678
AFAM_HFQNLGK_vs_IBP3_FLNVLSPR
38 и 99
0,028
0,636
ENPP2_TYLHTYESEI_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
83 и 135
0,018
0,647
AFAM_HFQNLGK_vs_IBP3_YGQPLPGYTTK
38 и 100
0,008
0,666
ENPP2_TYLHTYESEI_vs_PSG9_LFIPQITR
83 и 136
0,027
0,637
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,001
0,713
ENPP2_TYLHTYESEI_vs_SHBG_IALGGLLFPASNLR
83 и 18
0,000
0,722
AFAM_HFQNLGK_vs_PRG2_WNFAYWAAHQPWSR
38 и 129
0,036
0,631
ENPP2_TYLHTYESEI_vs_SOM2.CSH_NYGLLYCFR
83 и 138
0,019
0,646
AFAM_HFQNLGK_vs_SHBG_IALGGLLFPASNLR
38 и 18
0,035
0,631
ENPP2_TYLHTYESEI_vs_SOM2.CSH_SVEGSCGF
83 и 139
0,010
0,661
AFAM_HFQNLGK_vs_TENX_LNWEAPPGAFDSFLLR
38 и 141
0,008
0,664
ENPP2_TYLHTYESEI_vs_SPRL1_VLTHSELAPLR
83 и 140
0,003
0,686
AFAM_HFQNLGK_vs_TENX_LSQLSVTDVTTSSLR
38 и 142
0,023
0,641
ENPP2_TYLHTYESEI_vs_TENX_LNWEAPPGAFDSFLLR
83 и 141
0,000
0,728
ANGT_DPTFIPAPIQAK_vs_TENX_LNWEAPPGAFDSFLLR
42 и 141
0,020
0,644
ENPP2_TYLHTYESEI_vs_TENX_LSQLSVTDVTTSSLR
83 и 142
0,001
0,712
APOC3_GWVTDGFSSLK_vs_ALS_IRPHTFTGLSGLR
47 и 40
0,014
0,653
ENPP2_TYLHTYESEI_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
83 и 144
0,003
0,685
APOC3_GWVTDGFSSLK_vs_C163A_INPASLDK
47 и 54
0,045
0,625
ENPP2_TYLHTYESEI_vs_VTDB_ELPEHTVK
83 и 147
0,001
0,713
APOC3_GWVTDGFSSLK_vs_CHL1_VIAVNEVGR
47 и 66
0,010
0,660
FBLN3_IPSNPSHR_vs_SHBG_IALGGLLFPASNLR
87 и 18
0,041
0,627
APOC3_GWVTDGFSSLK_vs_CRIS3_AVSPPAR
47 и 78
0,006
0,669
FETUA_FSVVYAK_vs_CHL1_VIAVNEVGR
88 и 66
0,016
0,650
APOC3_GWVTDGFSSLK_vs_CRIS3_YEDLYSNCK
47 и 79
0,006
0,670
FETUA_FSVVYAK_vs_CRIS3_AVSPPAR
88 и 78
0,030
0,635
APOC3_GWVTDGFSSLK_vs_CSH_AHQLAIDTYQEFEETYIPK
47 и 80
0,013
0,654
FETUA_FSVVYAK_vs_CRIS3_YEDLYSNCK
88 и 79
0,017
0,648
APOC3_GWVTDGFSSLK_vs_CSH_ISLLLIESWLEPVR
47 и 81
0,017
0,648
FETUA_FSVVYAK_vs_IBP3_FLNVLSPR
88 и 99
0,018
0,646
APOC3_GWVTDGFSSLK_vs_FBLN1_TGYYFDGISR
47 и 86
0,012
0,656
FETUA_FSVVYAK_vs_IBP3_YGQPLPGYTTK
88 и 100
0,014
0,652
APOC3_GWVTDGFSSLK_vs_IBP1_VVESLAK
47 и 97
0,033
0,633
FETUA_FSVVYAK_vs_IGF2_GIVEECCFR
88 и 103
0,001
0,701
APOC3_GWVTDGFSSLK_vs_IBP2_LIQGAPTIR
47 и 98
0,021
0,644
FETUA_FSVVYAK_vs_ITIH4_ILDDLSPR
88 и 112
0,034
0,632
APOC3_GWVTDGFSSLK_vs_IBP3_FLNVLSPR
47 и 99
0,003
0,685
FETUA_FSVVYAK_vs_LYAM1_SYYWIGIR
88 и 120
0,023
0,642
APOC3_GWVTDGFSSLK_vs_IBP3_YGQPLPGYTTK
47 и 100
0,003
0,684
FETUA_FSVVYAK_vs_PGRP2_AGLLRPDYALLGHR
88 и 126
0,011
0,659
APOC3_GWVTDGFSSLK_vs_IGF2_GIVEECCFR
47 и 103
0,001
0,699
FETUA_FSVVYAK_vs_PRG2_WNFAYWAAHQPWSR
88 и 129
0,012
0,657
APOC3_GWVTDGFSSLK_vs_ITIH4_ILDDLSPR
47 и 112
0,003
0,682
FETUA_FSVVYAK_vs_PSG3_VSAPSGTGHLPGLNPL
88 и 134
0,033
0,633
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,002
0,692
FETUA_FSVVYAK_vs_SHBG_IALGGLLFPASNLR
88 и 18
0,010
0,660
APOC3_GWVTDGFSSLK_vs_NCAM1_GLGEISAASEFK
47 и 121
0,016
0,650
FETUA_FSVVYAK_vs_TENX_LNWEAPPGAFDSFLLR
88 и 141
0,002
0,694
APOC3_GWVTDGFSSLK_vs_PGRP2_AGLLRPDYALLGHR
47 и 126
0,001
0,699
FETUA_FSVVYAK_vs_TENX_LSQLSVTDVTTSSLR
88 и 142
0,007
0,668
APOC3_GWVTDGFSSLK_vs_PRG2_WNFAYWAAHQPWSR
47 и 129
0,006
0,670
FETUA_FSVVYAK_vs_VTDB_ELPEHTVK
88 и 147
0,007
0,668
APOC3_GWVTDGFSSLK_vs_PSG3_VSAPSGTGHLPGLNPL
47 и 134
0,010
0,660
FETUA_HTLNQIDEVK_vs_CHL1_VIAVNEVGR
89 и 66
0,013
0,654
APOC3_GWVTDGFSSLK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
47 и 135
0,018
0,647
FETUA_HTLNQIDEVK_vs_CRIS3_AVSPPAR
89 и 78
0,046
0,624
APOC3_GWVTDGFSSLK_vs_PSG9_LFIPQITR
47 и 136
0,018
0,647
FETUA_HTLNQIDEVK_vs_CRIS3_YEDLYSNCK
89 и 79
0,027
0,638
APOC3_GWVTDGFSSLK_vs_SHBG_IALGGLLFPASNLR
47 и 18
0,002
0,688
FETUA_HTLNQIDEVK_vs_IBP3_FLNVLSPR
89 и 99
0,036
0,630
APOC3_GWVTDGFSSLK_vs_SOM2.CSH_NYGLLYCFR
47 и 138
0,028
0,636
FETUA_HTLNQIDEVK_vs_IBP3_YGQPLPGYTTK
89 и 100
0,041
0,627
APOC3_GWVTDGFSSLK_vs_SOM2.CSH_SVEGSCGF
47 и 139
0,011
0,658
FETUA_HTLNQIDEVK_vs_IGF2_GIVEECCFR
89 и 103
0,004
0,681
APOC3_GWVTDGFSSLK_vs_SPRL1_VLTHSELAPLR
47 и 140
0,008
0,665
FETUA_HTLNQIDEVK_vs_LYAM1_SYYWIGIR
89 и 120
0,041
0,627
APOC3_GWVTDGFSSLK_vs_TENX_LNWEAPPGAFDSFLLR
47 и 141
0,002
0,693
FETUA_HTLNQIDEVK_vs_PGRP2_AGLLRPDYALLGHR
89 и 126
0,024
0,640
APOC3_GWVTDGFSSLK_vs_TENX_LSQLSVTDVTTSSLR
47 и 142
0,003
0,682
FETUA_HTLNQIDEVK_vs_PRG2_WNFAYWAAHQPWSR
89 и 129
0,015
0,651
APOC3_GWVTDGFSSLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
47 и 144
0,013
0,654
FETUA_HTLNQIDEVK_vs_PSG3_VSAPSGTGHLPGLNPL
89 и 134
0,037
0,630
APOC3_GWVTDGFSSLK_vs_VTDB_ELPEHTVK
47 и 147
0,003
0,683
FETUA_HTLNQIDEVK_vs_SHBG_IALGGLLFPASNLR
89 и 18
0,009
0,662
APOH_ATVVYQGER_vs_TENX_LNWEAPPGAFDSFLLR
48 и 141
0,042
0,627
FETUA_HTLNQIDEVK_vs_TENX_LNWEAPPGAFDSFLLR
89 и 141
0,001
0,701
B2MG_VEHSDLSFSK_vs_IGF2_GIVEECCFR
50 и 103
0,026
0,638
FETUA_HTLNQIDEVK_vs_TENX_LSQLSVTDVTTSSLR
89 и 142
0,005
0,673
B2MG_VEHSDLSFSK_vs_LYAM1_SYYWIGIR
50 и 120
0,037
0,630
FETUA_HTLNQIDEVK_vs_VTDB_ELPEHTVK
89 и 147
0,025
0,639
B2MG_VEHSDLSFSK_vs_PGRP2_AGLLRPDYALLGHR
50 и 126
0,033
0,633
HABP2_FLNWIK_vs_IBP3_FLNVLSPR
92 и 99
0,039
0,628
B2MG_VEHSDLSFSK_vs_SHBG_IALGGLLFPASNLR
50 и 18
0,021
0,644
HABP2_FLNWIK_vs_IBP3_YGQPLPGYTTK
92 и 100
0,039
0,629
B2MG_VEHSDLSFSK_vs_TENX_LNWEAPPGAFDSFLLR
50 и 141
0,008
0,664
HABP2_FLNWIK_vs_IGF2_GIVEECCFR
92 и 103
0,005
0,674
B2MG_VEHSDLSFSK_vs_TENX_LSQLSVTDVTTSSLR
50 и 142
0,042
0,627
HABP2_FLNWIK_vs_SHBG_IALGGLLFPASNLR
92 и 18
0,046
0,624
B2MG_VNHVTLSQPK_vs_CRIS3_YEDLYSNCK
51 и 79
0,046
0,624
HABP2_FLNWIK_vs_TENX_LNWEAPPGAFDSFLLR
92 и 141
0,006
0,669
B2MG_VNHVTLSQPK_vs_IGF2_GIVEECCFR
51 и 103
0,006
0,672
HABP2_FLNWIK_vs_TENX_LSQLSVTDVTTSSLR
92 и 142
0,021
0,644
B2MG_VNHVTLSQPK_vs_ITIH4_ILDDLSPR
51 и 112
0,038
0,629
HEMO_NFPSPVDAAFR_vs_IGF2_GIVEECCFR
93 и 103
0,043
0,626
B2MG_VNHVTLSQPK_vs_LYAM1_SYYWIGIR
51 и 120
0,017
0,648
HEMO_NFPSPVDAAFR_vs_PGRP2_AGLLRPDYALLGHR
93 и 126
0,050
0,622
B2MG_VNHVTLSQPK_vs_PGRP2_AGLLRPDYALLGHR
51 и 126
0,012
0,657
HEMO_NFPSPVDAAFR_vs_SHBG_IALGGLLFPASNLR
93 и 18
0,017
0,648
B2MG_VNHVTLSQPK_vs_PRG2_WNFAYWAAHQPWSR
51 и 129
0,050
0,622
HEMO_NFPSPVDAAFR_vs_TENX_LNWEAPPGAFDSFLLR
93 и 141
0,013
0,654
B2MG_VNHVTLSQPK_vs_SHBG_IALGGLLFPASNLR
51 и 18
0,009
0,662
IBP4_QCHPALDGQR_vs_CHL1_VIAVNEVGR
2 и 66
0,023
0,642
B2MG_VNHVTLSQPK_vs_TENX_LNWEAPPGAFDSFLLR
51 и 141
0,005
0,675
IBP4_QCHPALDGQR_vs_CRIS3_AVSPPAR
2 и 78
0,027
0,638
B2MG_VNHVTLSQPK_vs_TENX_LSQLSVTDVTTSSLR
51 и 142
0,030
0,635
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,013
0,655
BGH3_LTLLAPLNSVFK_vs_CRIS3_YEDLYSNCK
52 и 79
0,023
0,641
IBP4_QCHPALDGQR_vs_IGF2_GIVEECCFR
2 и 103
0,005
0,676
BGH3_LTLLAPLNSVFK_vs_IGF2_GIVEECCFR
52 и 103
0,009
0,662
IBP4_QCHPALDGQR_vs_ITIH4_ILDDLSPR
2 и 112
0,014
0,653
BGH3_LTLLAPLNSVFK_vs_LYAM1_SYYWIGIR
52 и 120
0,048
0,623
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,013
0,654
BGH3_LTLLAPLNSVFK_vs_PGRP2_AGLLRPDYALLGHR
52 и 126
0,050
0,622
IBP4_QCHPALDGQR_vs_PGRP2_AGLLRPDYALLGHR
2 и 126
0,005
0,674
BGH3_LTLLAPLNSVFK_vs_PRG2_WNFAYWAAHQPWSR
52 и 129
0,043
0,626
IBP4_QCHPALDGQR_vs_PRG2_WNFAYWAAHQPWSR
2 и 129
0,016
0,649
BGH3_LTLLAPLNSVFK_vs_SHBG_IALGGLLFPASNLR
52 и 18
0,045
0,625
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,003
0,685
BGH3_LTLLAPLNSVFK_vs_TENX_LNWEAPPGAFDSFLLR
52 и 141
0,011
0,658
IBP4_QCHPALDGQR_vs_TENX_LNWEAPPGAFDSFLLR
2 и 141
0,001
0,702
BGH3_LTLLAPLNSVFK_vs_TENX_LSQLSVTDVTTSSLR
52 и 142
0,039
0,628
IBP4_QCHPALDGQR_vs_TENX_LSQLSVTDVTTSSLR
2 и 142
0,016
0,649
C1QB_VPGLYYFTYHASSR_vs_IGF2_GIVEECCFR
55 и 103
0,014
0,652
IBP4_QCHPALDGQR_vs_VTDB_ELPEHTVK
2 и 147
0,022
0,643
C1QB_VPGLYYFTYHASSR_vs_PRG2_WNFAYWAAHQPWSR
55 и 129
0,049
0,623
INHBC_LDFHFSSDR_vs_CHL1_VIAVNEVGR
107 и 66
0,016
0,649
C1QB_VPGLYYFTYHASSR_vs_SHBG_IALGGLLFPASNLR
55 и 18
0,040
0,628
INHBC_LDFHFSSDR_vs_CRIS3_AVSPPAR
107 и 78
0,044
0,625
C1QB_VPGLYYFTYHASSR_vs_TENX_LNWEAPPGAFDSFLLR
55 и 141
0,009
0,663
INHBC_LDFHFSSDR_vs_CRIS3_YEDLYSNCK
107 и 79
0,025
0,639
C1QB_VPGLYYFTYHASSR_vs_TENX_LSQLSVTDVTTSSLR
55 и 142
0,038
0,629
INHBC_LDFHFSSDR_vs_IBP3_FLNVLSPR
107 и 99
0,014
0,653
CATD_VGFAEAAR_vs_ALS_IRPHTFTGLSGLR
57 и 40
0,024
0,641
INHBC_LDFHFSSDR_vs_IBP3_YGQPLPGYTTK
107 и 100
0,012
0,655
CATD_VGFAEAAR_vs_CHL1_VIAVNEVGR
57 и 66
0,009
0,662
INHBC_LDFHFSSDR_vs_IGF2_GIVEECCFR
107 и 103
0,001
0,707
CATD_VGFAEAAR_vs_CRIS3_AVSPPAR
57 и 78
0,014
0,652
INHBC_LDFHFSSDR_vs_ITIH4_ILDDLSPR
107 и 112
0,009
0,662
CATD_VGFAEAAR_vs_CRIS3_YEDLYSNCK
57 и 79
0,008
0,666
INHBC_LDFHFSSDR_vs_LYAM1_SYYWIGIR
107 и 120
0,014
0,652
CATD_VGFAEAAR_vs_CSH_AHQLAIDTYQEFEETYIPK
57 и 80
0,038
0,629
INHBC_LDFHFSSDR_vs_PGRP2_AGLLRPDYALLGHR
107 и 126
0,006
0,672
CATD_VGFAEAAR_vs_CSH_ISLLLIESWLEPVR
57 и 81
0,048
0,623
INHBC_LDFHFSSDR_vs_PRG2_WNFAYWAAHQPWSR
107 и 129
0,031
0,634
CATD_VGFAEAAR_vs_FBLN1_TGYYFDGISR
57 и 86
0,040
0,628
INHBC_LDFHFSSDR_vs_PSG3_VSAPSGTGHLPGLNPL
107 и 134
0,019
0,646
CATD_VGFAEAAR_vs_IBP2_LIQGAPTIR
57 и 98
0,049
0,623
INHBC_LDFHFSSDR_vs_SHBG_IALGGLLFPASNLR
107 и 18
0,006
0,672
CATD_VGFAEAAR_vs_IBP3_FLNVLSPR
57 и 99
0,006
0,671
INHBC_LDFHFSSDR_vs_SPRL1_VLTHSELAPLR
107 и 140
0,035
0,631
CATD_VGFAEAAR_vs_IBP3_YGQPLPGYTTK
57 и 100
0,007
0,667
INHBC_LDFHFSSDR_vs_TENX_LNWEAPPGAFDSFLLR
107 и 141
0,003
0,684
CATD_VGFAEAAR_vs_IGF2_GIVEECCFR
57 и 103
0,001
0,714
INHBC_LDFHFSSDR_vs_TENX_LSQLSVTDVTTSSLR
107 и 142
0,009
0,662
CATD_VGFAEAAR_vs_ITIH4_ILDDLSPR
57 и 112
0,010
0,661
INHBC_LDFHFSSDR_vs_VTDB_ELPEHTVK
107 и 147
0,008
0,666
CATD_VGFAEAAR_vs_LYAM1_SYYWIGIR
57 и 120
0,006
0,671
ITIH3_ALDLSLK_vs_CRIS3_YEDLYSNCK
111 и 79
0,043
0,626
CATD_VGFAEAAR_vs_NCAM1_GLGEISAASEFK
57 и 121
0,026
0,639
ITIH3_ALDLSLK_vs_IGF2_GIVEECCFR
111 и 103
0,037
0,630
CATD_VGFAEAAR_vs_PGRP2_AGLLRPDYALLGHR
57 и 126
0,003
0,685
ITIH3_ALDLSLK_vs_LYAM1_SYYWIGIR
111 и 120
0,034
0,632
CATD_VGFAEAAR_vs_PRG2_WNFAYWAAHQPWSR
57 и 129
0,002
0,688
ITIH3_ALDLSLK_vs_PGRP2_AGLLRPDYALLGHR
111 и 126
0,008
0,665
CATD_VGFAEAAR_vs_PSG3_VSAPSGTGHLPGLNPL
57 и 134
0,008
0,666
ITIH3_ALDLSLK_vs_PRG2_WNFAYWAAHQPWSR
111 и 129
0,035
0,631
CATD_VGFAEAAR_vs_SHBG_IALGGLLFPASNLR
57 и 18
0,009
0,662
ITIH3_ALDLSLK_vs_SHBG_IALGGLLFPASNLR
111 и 18
0,010
0,660
CATD_VGFAEAAR_vs_SOM2.CSH_SVEGSCGF
57 и 139
0,039
0,629
ITIH3_ALDLSLK_vs_TENX_LNWEAPPGAFDSFLLR
111 и 141
0,016
0,650
CATD_VGFAEAAR_vs_SPRL1_VLTHSELAPLR
57 и 140
0,012
0,656
ITIH3_ALDLSLK_vs_TENX_LSQLSVTDVTTSSLR
111 и 142
0,049
0,622
CATD_VGFAEAAR_vs_TENX_LNWEAPPGAFDSFLLR
57 и 141
0,000
0,739
KNG1_DIPTNSPELEETLTHTITK_vs_IGF2_GIVEECCFR
116 и 103
0,026
0,639
CATD_VGFAEAAR_vs_TENX_LSQLSVTDVTTSSLR
57 и 142
0,001
0,713
KNG1_DIPTNSPELEETLTHTITK_vs_SHBG_IALGGLLFPASNLR
116 и 18
0,023
0,642
CATD_VGFAEAAR_vs_VTDB_ELPEHTVK
57 и 147
0,008
0,666
KNG1_DIPTNSPELEETLTHTITK_vs_TENX_LNWEAPPGAFDSFLLR
116 и 141
0,006
0,671
CATD_VSTLPAITLK_vs_ALS_IRPHTFTGLSGLR
58 и 40
0,037
0,630
KNG1_DIPTNSPELEETLTHTITK_vs_TENX_LSQLSVTDVTTSSLR
116 и 142
0,032
0,634
CATD_VSTLPAITLK_vs_CHL1_VIAVNEVGR
58 и 66
0,009
0,663
KNG1_QVVAGLNFR_vs_CHL1_VIAVNEVGR
117 и 66
0,037
0,630
CATD_VSTLPAITLK_vs_CRIS3_AVSPPAR
58 и 78
0,022
0,643
KNG1_QVVAGLNFR_vs_CRIS3_YEDLYSNCK
117 и 79
0,049
0,622
CATD_VSTLPAITLK_vs_CRIS3_YEDLYSNCK
58 и 79
0,013
0,655
KNG1_QVVAGLNFR_vs_IGF2_GIVEECCFR
117 и 103
0,014
0,653
CATD_VSTLPAITLK_vs_CSH_ISLLLIESWLEPVR
58 и 81
0,049
0,623
KNG1_QVVAGLNFR_vs_ITIH4_ILDDLSPR
117 и 112
0,026
0,638
CATD_VSTLPAITLK_vs_FBLN1_TGYYFDGISR
58 и 86
0,034
0,632
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,024
0,641
CATD_VSTLPAITLK_vs_IBP2_LIQGAPTIR
58 и 98
0,037
0,630
KNG1_QVVAGLNFR_vs_PGRP2_AGLLRPDYALLGHR
117 и 126
0,016
0,649
CATD_VSTLPAITLK_vs_IBP3_FLNVLSPR
58 и 99
0,006
0,671
KNG1_QVVAGLNFR_vs_PRG2_WNFAYWAAHQPWSR
117 и 129
0,037
0,630
CATD_VSTLPAITLK_vs_IBP3_YGQPLPGYTTK
58 и 100
0,010
0,660
KNG1_QVVAGLNFR_vs_SHBG_IALGGLLFPASNLR
117 и 18
0,005
0,675
CATD_VSTLPAITLK_vs_IGF2_GIVEECCFR
58 и 103
0,001
0,712
KNG1_QVVAGLNFR_vs_TENX_LNWEAPPGAFDSFLLR
117 и 141
0,003
0,685
CATD_VSTLPAITLK_vs_ITIH4_ILDDLSPR
58 и 112
0,014
0,653
KNG1_QVVAGLNFR_vs_TENX_LSQLSVTDVTTSSLR
117 и 142
0,032
0,634
CATD_VSTLPAITLK_vs_LYAM1_SYYWIGIR
58 и 120
0,011
0,659
KNG1_QVVAGLNFR_vs_VTDB_ELPEHTVK
117 и 147
0,030
0,635
CATD_VSTLPAITLK_vs_NCAM1_GLGEISAASEFK
58 и 121
0,031
0,634
LBP_ITGFLKPGK_vs_CHL1_VIAVNEVGR
118 и 66
0,029
0,636
CATD_VSTLPAITLK_vs_PGRP2_AGLLRPDYALLGHR
58 и 126
0,002
0,691
LBP_ITGFLKPGK_vs_CRIS3_AVSPPAR
118 и 78
0,009
0,663
CATD_VSTLPAITLK_vs_PRG2_WNFAYWAAHQPWSR
58 и 129
0,005
0,677
LBP_ITGFLKPGK_vs_CRIS3_YEDLYSNCK
118 и 79
0,005
0,674
CATD_VSTLPAITLK_vs_PSG3_VSAPSGTGHLPGLNPL
58 и 134
0,012
0,657
LBP_ITGFLKPGK_vs_IBP2_LIQGAPTIR
118 и 98
0,035
0,631
CATD_VSTLPAITLK_vs_SHBG_IALGGLLFPASNLR
58 и 18
0,005
0,677
LBP_ITGFLKPGK_vs_IBP3_FLNVLSPR
118 и 99
0,022
0,642
CATD_VSTLPAITLK_vs_SPRL1_VLTHSELAPLR
58 и 140
0,028
0,637
LBP_ITGFLKPGK_vs_IBP3_YGQPLPGYTTK
118 и 100
0,012
0,657
CATD_VSTLPAITLK_vs_TENX_LNWEAPPGAFDSFLLR
58 и 141
0,000
0,731
LBP_ITGFLKPGK_vs_IGF2_GIVEECCFR
118 и 103
0,003
0,684
CATD_VSTLPAITLK_vs_TENX_LSQLSVTDVTTSSLR
58 и 142
0,001
0,710
LBP_ITGFLKPGK_vs_ITIH4_ILDDLSPR
118 и 112
0,020
0,645
CATD_VSTLPAITLK_vs_VTDB_ELPEHTVK
58 и 147
0,019
0,646
LBP_ITGFLKPGK_vs_LYAM1_SYYWIGIR
118 и 120
0,008
0,664
CBPN_EALIQFLEQVHQGIK_vs_SHBG_IALGGLLFPASNLR
59 и 18
0,037
0,630
LBP_ITGFLKPGK_vs_PGRP2_AGLLRPDYALLGHR
118 и 126
0,014
0,653
CBPN_EALIQFLEQVHQGIK_vs_TENX_LNWEAPPGAFDSFLLR
59 и 141
0,027
0,637
LBP_ITGFLKPGK_vs_PRG2_WNFAYWAAHQPWSR
118 и 129
0,018
0,646
CBPN_NNANGVDLNR_vs_IGF2_GIVEECCFR
60 и 103
0,030
0,635
LBP_ITGFLKPGK_vs_PSG3_VSAPSGTGHLPGLNPL
118 и 134
0,016
0,650
CBPN_NNANGVDLNR_vs_PGRP2_AGLLRPDYALLGHR
60 и 126
0,041
0,627
LBP_ITGFLKPGK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
118 и 135
0,045
0,625
CBPN_NNANGVDLNR_vs_SHBG_IALGGLLFPASNLR
60 и 18
0,024
0,641
LBP_ITGFLKPGK_vs_SHBG_IALGGLLFPASNLR
118 и 18
0,009
0,662
CBPN_NNANGVDLNR_vs_TENX_LNWEAPPGAFDSFLLR
60 и 141
0,011
0,657
LBP_ITGFLKPGK_vs_SPRL1_VLTHSELAPLR
118 и 140
0,024
0,640
CBPN_NNANGVDLNR_vs_TENX_LSQLSVTDVTTSSLR
60 и 142
0,031
0,634
LBP_ITGFLKPGK_vs_TENX_LNWEAPPGAFDSFLLR
118 и 141
0,005
0,676
CD14_LTVGAAQVPAQLLVGALR_vs_IGF2_GIVEECCFR
61 и 103
0,028
0,636
LBP_ITGFLKPGK_vs_TENX_LSQLSVTDVTTSSLR
118 и 142
0,019
0,646
CD14_LTVGAAQVPAQLLVGALR_vs_SHBG_IALGGLLFPASNLR
61 и 18
0,033
0,632
LBP_ITGFLKPGK_vs_VTDB_ELPEHTVK
118 и 147
0,010
0,659
CD14_LTVGAAQVPAQLLVGALR_vs_TENX_LNWEAPPGAFDSFLLR
61 и 141
0,014
0,652
LBP_ITLPDFTGDLR_vs_ALS_IRPHTFTGLSGLR
119 и 40
0,019
0,646
CD14_SWLAELQQWLKPGLK_vs_SHBG_IALGGLLFPASNLR
62 и 18
0,039
0,628
LBP_ITLPDFTGDLR_vs_CHL1_VIAVNEVGR
119 и 66
0,009
0,663
CD14_SWLAELQQWLKPGLK_vs_TENX_LNWEAPPGAFDSFLLR
62 и 141
0,019
0,646
LBP_ITLPDFTGDLR_vs_CRIS3_AVSPPAR
119 и 78
0,003
0,686
CFAB_YGLVTYATYPK_vs_IGF2_GIVEECCFR
64 и 103
0,010
0,660
LBP_ITLPDFTGDLR_vs_CRIS3_YEDLYSNCK
119 и 79
0,002
0,696
CFAB_YGLVTYATYPK_vs_PGRP2_AGLLRPDYALLGHR
64 и 126
0,044
0,625
LBP_ITLPDFTGDLR_vs_IBP1_VVESLAK
119 и 97
0,046
0,624
CFAB_YGLVTYATYPK_vs_SHBG_IALGGLLFPASNLR
64 и 18
0,049
0,623
LBP_ITLPDFTGDLR_vs_IBP2_LIQGAPTIR
119 и 98
0,012
0,656
CFAB_YGLVTYATYPK_vs_TENX_LNWEAPPGAFDSFLLR
64 и 141
0,015
0,651
LBP_ITLPDFTGDLR_vs_IBP3_FLNVLSPR
119 и 99
0,004
0,679
CFAB_YGLVTYATYPK_vs_TENX_LSQLSVTDVTTSSLR
64 и 142
0,047
0,623
LBP_ITLPDFTGDLR_vs_IBP3_YGQPLPGYTTK
119 и 100
0,002
0,694
CO5_TLLPVSKPEIR_vs_IGF2_GIVEECCFR
70 и 103
0,047
0,623
LBP_ITLPDFTGDLR_vs_IGF2_GIVEECCFR
119 и 103
0,001
0,713
CO5_TLLPVSKPEIR_vs_SHBG_IALGGLLFPASNLR
70 и 18
0,041
0,627
LBP_ITLPDFTGDLR_vs_ITIH4_ILDDLSPR
119 и 112
0,003
0,688
CO5_TLLPVSKPEIR_vs_TENX_LNWEAPPGAFDSFLLR
70 и 141
0,021
0,644
LBP_ITLPDFTGDLR_vs_LYAM1_SYYWIGIR
119 и 120
0,002
0,690
CO5_VFQFLEK_vs_IGF2_GIVEECCFR
71 и 103
0,041
0,627
LBP_ITLPDFTGDLR_vs_NCAM1_GLGEISAASEFK
119 и 121
0,040
0,628
CO5_VFQFLEK_vs_SHBG_IALGGLLFPASNLR
71 и 18
0,042
0,627
LBP_ITLPDFTGDLR_vs_PGRP2_AGLLRPDYALLGHR
119 и 126
0,002
0,692
CO5_VFQFLEK_vs_TENX_LNWEAPPGAFDSFLLR
71 и 141
0,014
0,653
LBP_ITLPDFTGDLR_vs_PRG2_WNFAYWAAHQPWSR
119 и 129
0,007
0,667
CO5_VFQFLEK_vs_TENX_LSQLSVTDVTTSSLR
71 и 142
0,047
0,623
LBP_ITLPDFTGDLR_vs_PSG3_VSAPSGTGHLPGLNPL
119 и 134
0,007
0,667
CO6_ALNHLPLEYNSALYSR_vs_TENX_LNWEAPPGAFDSFLLR
72 и 141
0,043
0,626
LBP_ITLPDFTGDLR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
119 и 135
0,025
0,639
CO8A_SLLQPNK_vs_IGF2_GIVEECCFR
74 и 103
0,012
0,655
LBP_ITLPDFTGDLR_vs_PSG9_LFIPQITR
119 и 136
0,037
0,630
CO8A_SLLQPNK_vs_PGRP2_AGLLRPDYALLGHR
74 и 126
0,032
0,633
LBP_ITLPDFTGDLR_vs_SHBG_IALGGLLFPASNLR
119 и 18
0,002
0,690
CO8A_SLLQPNK_vs_SHBG_IALGGLLFPASNLR
74 и 18
0,048
0,623
LBP_ITLPDFTGDLR_vs_SOM2.CSH_SVEGSCGF
119 и 139
0,032
0,634
CO8A_SLLQPNK_vs_TENX_LNWEAPPGAFDSFLLR
74 и 141
0,009
0,662
LBP_ITLPDFTGDLR_vs_SPRL1_VLTHSELAPLR
119 и 140
0,007
0,668
CO8A_SLLQPNK_vs_TENX_LSQLSVTDVTTSSLR
74 и 142
0,021
0,644
LBP_ITLPDFTGDLR_vs_TENX_LNWEAPPGAFDSFLLR
119 и 141
0,001
0,705
CO8B_QALEEFQK_vs_IGF2_GIVEECCFR
76 и 103
0,009
0,663
LBP_ITLPDFTGDLR_vs_TENX_LSQLSVTDVTTSSLR
119 и 142
0,004
0,678
CO8B_QALEEFQK_vs_PGRP2_AGLLRPDYALLGHR
76 и 126
0,040
0,628
LBP_ITLPDFTGDLR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
119 и 144
0,020
0,645
CO8B_QALEEFQK_vs_SHBG_IALGGLLFPASNLR
76 и 18
0,045
0,625
LBP_ITLPDFTGDLR_vs_VTDB_ELPEHTVK
119 и 147
0,002
0,695
CO8B_QALEEFQK_vs_TENX_LNWEAPPGAFDSFLLR
76 и 141
0,006
0,670
PAPP1_DIPHWLNPTR_vs_C163A_INPASLDK
122 и 54
0,050
0,622
CO8B_QALEEFQK_vs_TENX_LSQLSVTDVTTSSLR
76 и 142
0,012
0,655
PAPP1_DIPHWLNPTR_vs_CSH_AHQLAIDTYQEFEETYIPK
122 и 80
0,035
0,631
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_ALS_IRPHTFTGLSGLR
82 и 40
0,002
0,695
PAPP1_DIPHWLNPTR_vs_CSH_ISLLLIESWLEPVR
122 и 81
0,044
0,625
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_C163A_INPASLDK
82 и 54
0,036
0,631
PAPP1_DIPHWLNPTR_vs_SOM2.CSH_NYGLLYCFR
122 и 138
0,021
0,643
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_CHL1_VIAVNEVGR
82 и 66
0,002
0,690
PEDF_LQSLFDSPDFSK_vs_CRIS3_YEDLYSNCK
124 и 79
0,030
0,635
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_CRIS3_AVSPPAR
82 и 78
0,005
0,673
PEDF_LQSLFDSPDFSK_vs_IGF2_GIVEECCFR
124 и 103
0,011
0,658
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_CRIS3_YEDLYSNCK
82 и 79
0,004
0,679
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,036
0,631
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_CSH_AHQLAIDTYQEFEETYIPK
82 и 80
0,012
0,656
PEDF_LQSLFDSPDFSK_vs_PGRP2_AGLLRPDYALLGHR
124 и 126
0,033
0,632
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_CSH_ISLLLIESWLEPVR
82 и 81
0,014
0,652
PEDF_LQSLFDSPDFSK_vs_TENX_LNWEAPPGAFDSFLLR
124 и 141
0,008
0,666
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_FBLN1_TGYYFDGISR
82 и 86
0,007
0,669
PEDF_LQSLFDSPDFSK_vs_TENX_LSQLSVTDVTTSSLR
124 и 142
0,035
0,631
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_IBP1_VVESLAK
82 и 97
0,027
0,638
PEDF_TVQAVLTVPK_vs_IGF2_GIVEECCFR
125 и 103
0,016
0,650
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_IBP2_LIQGAPTIR
82 и 98
0,008
0,664
PEDF_TVQAVLTVPK_vs_TENX_LNWEAPPGAFDSFLLR
125 и 141
0,016
0,650
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_IBP3_FLNVLSPR
82 и 99
0,001
0,708
PSG11_LFIPQITPK_vs_PRG2_WNFAYWAAHQPWSR
132 и 129
0,043
0,626
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_IBP3_YGQPLPGYTTK
82 и 100
0,001
0,708
PTGDS_GPGEDFR_vs_SHBG_IALGGLLFPASNLR
137 и 18
0,033
0,632
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_IGF2_GIVEECCFR
82 и 103
0,000
0,736
PTGDS_GPGEDFR_vs_TENX_LNWEAPPGAFDSFLLR
137 и 141
0,015
0,651
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_ITIH4_ILDDLSPR
82 и 112
0,001
0,710
PTGDS_GPGEDFR_vs_TENX_LSQLSVTDVTTSSLR
137 и 142
0,046
0,624
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_LYAM1_SYYWIGIR
82 и 120
0,001
0,704
THBG_AVLHIGEK_vs_TENX_LNWEAPPGAFDSFLLR
143 и 141
0,040
0,628
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_NCAM1_GLGEISAASEFK
82 и 121
0,013
0,655
VTNC_GQYCYELDEK_vs_CHL1_VIAVNEVGR
149 и 66
0,038
0,629
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PGRP2_AGLLRPDYALLGHR
82 и 126
0,001
0,708
VTNC_GQYCYELDEK_vs_CRIS3_YEDLYSNCK
149 и 79
0,049
0,623
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PRG2_WNFAYWAAHQPWSR
82 и 129
0,003
0,684
VTNC_GQYCYELDEK_vs_IBP3_FLNVLSPR
149 и 99
0,017
0,648
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PSG3_VSAPSGTGHLPGLNPL
82 и 134
0,004
0,681
VTNC_GQYCYELDEK_vs_IBP3_YGQPLPGYTTK
149 и 100
0,020
0,645
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
82 и 135
0,016
0,650
VTNC_GQYCYELDEK_vs_IGF2_GIVEECCFR
149 и 103
0,002
0,689
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PSG9_LFIPQITR
82 и 136
0,027
0,637
VTNC_GQYCYELDEK_vs_ITIH4_ILDDLSPR
149 и 112
0,024
0,641
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_SHBG_IALGGLLFPASNLR
82 и 18
0,000
0,720
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,039
0,629
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_SOM2.CSH_NYGLLYCFR
82 и 138
0,032
0,633
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,028
0,636
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_SOM2.CSH_SVEGSCGF
82 и 139
0,020
0,645
VTNC_GQYCYELDEK_vs_PRG2_WNFAYWAAHQPWSR
149 и 129
0,045
0,625
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_SPRL1_VLTHSELAPLR
82 и 140
0,005
0,674
VTNC_GQYCYELDEK_vs_PSG3_VSAPSGTGHLPGLNPL
149 и 134
0,039
0,629
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_TENX_LNWEAPPGAFDSFLLR
82 и 141
0,001
0,715
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,006
0,672
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_TENX_LSQLSVTDVTTSSLR
82 и 142
0,001
0,700
VTNC_GQYCYELDEK_vs_TENX_LNWEAPPGAFDSFLLR
149 и 141
0,003
0,683
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
82 и 144
0,006
0,671
VTNC_GQYCYELDEK_vs_TENX_LSQLSVTDVTTSSLR
149 и 142
0,013
0,654
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_VTDB_ELPEHTVK
82 и 147
0,001
0,711
VTNC_GQYCYELDEK_vs_VTDB_ELPEHTVK
149 и 147
0,005
0,673
ENPP2_TYLHTYESEI_vs_ALS_IRPHTFTGLSGLR
83 и 40
0,001
0,707
VTNC_VDTVDPPYPR_vs_CHL1_VIAVNEVGR
150 и 66
0,025
0,640
ENPP2_TYLHTYESEI_vs_C163A_INPASLDK
83 и 54
0,016
0,650
VTNC_VDTVDPPYPR_vs_CRIS3_AVSPPAR
150 и 78
0,050
0,622
ENPP2_TYLHTYESEI_vs_CHL1_VIAVNEVGR
83 и 66
0,002
0,694
VTNC_VDTVDPPYPR_vs_CRIS3_YEDLYSNCK
150 и 79
0,040
0,628
ENPP2_TYLHTYESEI_vs_CRIS3_AVSPPAR
83 и 78
0,004
0,680
VTNC_VDTVDPPYPR_vs_IBP3_FLNVLSPR
150 и 99
0,021
0,644
ENPP2_TYLHTYESEI_vs_CRIS3_YEDLYSNCK
83 и 79
0,002
0,689
VTNC_VDTVDPPYPR_vs_IBP3_YGQPLPGYTTK
150 и 100
0,023
0,641
ENPP2_TYLHTYESEI_vs_CSH_AHQLAIDTYQEFEETYIPK
83 и 80
0,006
0,670
VTNC_VDTVDPPYPR_vs_IGF2_GIVEECCFR
150 и 103
0,003
0,686
ENPP2_TYLHTYESEI_vs_CSH_ISLLLIESWLEPVR
83 и 81
0,008
0,664
VTNC_VDTVDPPYPR_vs_ITIH4_ILDDLSPR
150 и 112
0,017
0,648
ENPP2_TYLHTYESEI_vs_FBLN1_TGYYFDGISR
83 и 86
0,007
0,669
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,018
0,646
ENPP2_TYLHTYESEI_vs_IBP1_VVESLAK
83 и 97
0,028
0,636
VTNC_VDTVDPPYPR_vs_PGRP2_AGLLRPDYALLGHR
150 и 126
0,020
0,645
ENPP2_TYLHTYESEI_vs_IBP2_LIQGAPTIR
83 и 98
0,007
0,666
VTNC_VDTVDPPYPR_vs_PRG2_WNFAYWAAHQPWSR
150 и 129
0,034
0,632
ENPP2_TYLHTYESEI_vs_IBP3_FLNVLSPR
83 и 99
0,000
0,719
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,005
0,675
ENPP2_TYLHTYESEI_vs_IBP3_YGQPLPGYTTK
83 и 100
0,001
0,716
VTNC_VDTVDPPYPR_vs_TENX_LNWEAPPGAFDSFLLR
150 и 141
0,003
0,687
ENPP2_TYLHTYESEI_vs_IGF2_GIVEECCFR
83 и 103
0,000
0,741
VTNC_VDTVDPPYPR_vs_TENX_LSQLSVTDVTTSSLR
150 и 142
0,013
0,654
ENPP2_TYLHTYESEI_vs_ITIH4_ILDDLSPR
83 и 112
0,001
0,716
VTNC_VDTVDPPYPR_vs_VTDB_ELPEHTVK
150 и 147
0,003
0,686
Таблица 40: эффективность классификации по обращениям, недели 20 и 21.
AUROC обращения для недель беременности 20 и 21 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, с BMI-стратификацией (>22≤37).
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн.
ROC_ AUC
A2GL_DLLLPQPDLR_vs_IGF2_GIVEECCFR
34 и 103
0,022
0,667
ENPP2_TYLHTYESEI_vs_PGRP2_AGLLRPDYALLGHR
83 и 126
0,002
0,725
A2GL_DLLLPQPDLR_vs_LYAM1_SYYWIGIR
34 и 120
0,049
0,644
ENPP2_TYLHTYESEI_vs_PSG3_VSAPSGTGHLPGLNPL
83 и 134
0,025
0,664
A2GL_DLLLPQPDLR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
34 и 135
0,044
0,647
ENPP2_TYLHTYESEI_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
83 и 135
0,004
0,712
A2GL_DLLLPQPDLR_vs_PSG9_LFIPQITR
34 и 136
0,043
0,648
ENPP2_TYLHTYESEI_vs_PSG9_LFIPQITR
83 и 136
0,008
0,693
AFAM_DADPDTFFAK_vs_IBP3_FLNVLSPR
37 и 99
0,032
0,656
ENPP2_TYLHTYESEI_vs_SHBG_IALGGLLFPASNLR
83 и 18
0,001
0,747
AFAM_DADPDTFFAK_vs_IBP3_YGQPLPGYTTK
37 и 100
0,005
0,703
ENPP2_TYLHTYESEI_vs_SOM2.CSH_NYGLLYCFR
83 и 138
0,028
0,661
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,004
0,710
ENPP2_TYLHTYESEI_vs_SOM2.CSH_SVEGSCGF
83 и 139
0,003
0,716
AFAM_HFQNLGK_vs_ALS_IRPHTFTGLSGLR
38 и 40
0,026
0,662
ENPP2_TYLHTYESEI_vs_SPRL1_VLTHSELAPLR
83 и 140
0,009
0,691
AFAM_HFQNLGK_vs_CRIS3_YEDLYSNCK
38 и 79
0,038
0,651
ENPP2_TYLHTYESEI_vs_TENX_LNWEAPPGAFDSFLLR
83 и 141
0,006
0,702
AFAM_HFQNLGK_vs_IBP3_FLNVLSPR
38 и 99
0,003
0,718
ENPP2_TYLHTYESEI_vs_TENX_LSQLSVTDVTTSSLR
83 и 142
0,013
0,682
AFAM_HFQNLGK_vs_IBP3_YGQPLPGYTTK
38 и 100
0,000
0,757
ENPP2_TYLHTYESEI_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
83 и 144
0,011
0,686
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,000
0,775
ENPP2_TYLHTYESEI_vs_VTDB_ELPEHTVK
83 и 147
0,005
0,705
AFAM_HFQNLGK_vs_LYAM1_SYYWIGIR
38 и 120
0,045
0,646
FETUA_FSVVYAK_vs_CRIS3_AVSPPAR
88 и 78
0,025
0,664
AFAM_HFQNLGK_vs_PGRP2_AGLLRPDYALLGHR
38 и 126
0,025
0,664
FETUA_FSVVYAK_vs_CRIS3_YEDLYSNCK
88 и 79
0,009
0,692
AFAM_HFQNLGK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
38 и 135
0,028
0,660
FETUA_FSVVYAK_vs_IBP3_FLNVLSPR
88 и 99
0,016
0,676
ANGT_DPTFIPAPIQAK_vs_CRIS3_AVSPPAR
42 и 78
0,044
0,647
FETUA_FSVVYAK_vs_IBP3_YGQPLPGYTTK
88 и 100
0,006
0,700
ANGT_DPTFIPAPIQAK_vs_CRIS3_YEDLYSNCK
42 и 79
0,031
0,657
FETUA_FSVVYAK_vs_IGF2_GIVEECCFR
88 и 103
0,003
0,718
ANGT_DPTFIPAPIQAK_vs_IBP3_FLNVLSPR
42 и 99
0,037
0,653
FETUA_FSVVYAK_vs_LYAM1_SYYWIGIR
88 и 120
0,024
0,665
ANGT_DPTFIPAPIQAK_vs_IBP3_YGQPLPGYTTK
42 и 100
0,011
0,686
FETUA_FSVVYAK_vs_PGRP2_AGLLRPDYALLGHR
88 и 126
0,047
0,645
ANGT_DPTFIPAPIQAK_vs_IGF2_GIVEECCFR
42 и 103
0,007
0,697
FETUA_FSVVYAK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
88 и 135
0,026
0,663
ANGT_DPTFIPAPIQAK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
42 и 135
0,048
0,645
FETUA_FSVVYAK_vs_SHBG_IALGGLLFPASNLR
88 и 18
0,040
0,650
ANGT_DPTFIPAPIQAK_vs_SHBG_IALGGLLFPASNLR
42 и 18
0,038
0,651
FETUA_FSVVYAK_vs_VTDB_ELPEHTVK
88 и 147
0,049
0,644
APOC3_GWVTDGFSSLK_vs_CRIS3_AVSPPAR
47 и 78
0,024
0,665
FETUA_HTLNQIDEVK_vs_CRIS3_AVSPPAR
89 и 78
0,037
0,653
APOC3_GWVTDGFSSLK_vs_CRIS3_YEDLYSNCK
47 и 79
0,015
0,678
FETUA_HTLNQIDEVK_vs_CRIS3_YEDLYSNCK
89 и 79
0,015
0,678
APOC3_GWVTDGFSSLK_vs_IBP3_FLNVLSPR
47 и 99
0,029
0,659
FETUA_HTLNQIDEVK_vs_IBP3_FLNVLSPR
89 и 99
0,017
0,675
APOC3_GWVTDGFSSLK_vs_IBP3_YGQPLPGYTTK
47 и 100
0,019
0,672
FETUA_HTLNQIDEVK_vs_IBP3_YGQPLPGYTTK
89 и 100
0,007
0,696
APOC3_GWVTDGFSSLK_vs_IGF2_GIVEECCFR
47 и 103
0,016
0,675
FETUA_HTLNQIDEVK_vs_IGF2_GIVEECCFR
89 и 103
0,003
0,715
APOC3_GWVTDGFSSLK_vs_ITIH4_ILDDLSPR
47 и 112
0,029
0,659
FETUA_HTLNQIDEVK_vs_LYAM1_SYYWIGIR
89 и 120
0,041
0,649
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,021
0,669
FETUA_HTLNQIDEVK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
89 и 135
0,029
0,659
APOC3_GWVTDGFSSLK_vs_PGRP2_AGLLRPDYALLGHR
47 и 126
0,018
0,673
FETUA_HTLNQIDEVK_vs_SHBG_IALGGLLFPASNLR
89 и 18
0,029
0,659
APOC3_GWVTDGFSSLK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
47 и 135
0,013
0,681
HABP2_FLNWIK_vs_IBP3_FLNVLSPR
92 и 99
0,044
0,647
APOC3_GWVTDGFSSLK_vs_PSG9_LFIPQITR
47 и 136
0,017
0,675
HABP2_FLNWIK_vs_IBP3_YGQPLPGYTTK
92 и 100
0,024
0,664
APOC3_GWVTDGFSSLK_vs_SHBG_IALGGLLFPASNLR
47 и 18
0,020
0,670
HABP2_FLNWIK_vs_IGF2_GIVEECCFR
92 и 103
0,018
0,673
APOC3_GWVTDGFSSLK_vs_SOM2.CSH_SVEGSCGF
47 и 139
0,031
0,658
HEMO_NFPSPVDAAFR_vs_CRIS3_AVSPPAR
93 и 78
0,047
0,645
APOC3_GWVTDGFSSLK_vs_VTDB_ELPEHTVK
47 и 147
0,050
0,643
HEMO_NFPSPVDAAFR_vs_CRIS3_YEDLYSNCK
93 и 79
0,025
0,664
B2MG_VEHSDLSFSK_vs_CRIS3_AVSPPAR
50 и 78
0,049
0,644
HEMO_NFPSPVDAAFR_vs_IBP3_FLNVLSPR
93 и 99
0,045
0,646
B2MG_VEHSDLSFSK_vs_CRIS3_YEDLYSNCK
50 и 79
0,021
0,668
HEMO_NFPSPVDAAFR_vs_IBP3_YGQPLPGYTTK
93 и 100
0,030
0,659
B2MG_VEHSDLSFSK_vs_IBP3_YGQPLPGYTTK
50 и 100
0,049
0,644
HEMO_NFPSPVDAAFR_vs_IGF2_GIVEECCFR
93 и 103
0,014
0,679
B2MG_VEHSDLSFSK_vs_IGF2_GIVEECCFR
50 и 103
0,017
0,675
HEMO_NFPSPVDAAFR_vs_ITIH4_ILDDLSPR
93 и 112
0,017
0,674
B2MG_VEHSDLSFSK_vs_LYAM1_SYYWIGIR
50 и 120
0,024
0,665
HEMO_NFPSPVDAAFR_vs_LYAM1_SYYWIGIR
93 и 120
0,048
0,645
B2MG_VEHSDLSFSK_vs_PGRP2_AGLLRPDYALLGHR
50 и 126
0,043
0,648
HEMO_NFPSPVDAAFR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
93 и 135
0,031
0,658
B2MG_VEHSDLSFSK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
50 и 135
0,044
0,647
HEMO_NFPSPVDAAFR_vs_SHBG_IALGGLLFPASNLR
93 и 18
0,021
0,669
B2MG_VEHSDLSFSK_vs_SHBG_IALGGLLFPASNLR
50 и 18
0,037
0,652
HLACI_WAAVVVPSGEEQR_vs_CRIS3_YEDLYSNCK
95 и 79
0,047
0,645
B2MG_VNHVTLSQPK_vs_CRIS3_AVSPPAR
51 и 78
0,047
0,645
HLACI_WAAVVVPSGEEQR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
95 и 135
0,007
0,695
B2MG_VNHVTLSQPK_vs_CRIS3_YEDLYSNCK
51 и 79
0,028
0,661
HLACI_WAAVVVPSGEEQR_vs_PSG9_LFIPQITR
95 и 136
0,011
0,685
B2MG_VNHVTLSQPK_vs_IBP3_YGQPLPGYTTK
51 и 100
0,028
0,661
IBP4_QCHPALDGQR_vs_CRIS3_AVSPPAR
2 и 78
0,010
0,689
B2MG_VNHVTLSQPK_vs_IGF2_GIVEECCFR
51 и 103
0,008
0,695
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,002
0,722
B2MG_VNHVTLSQPK_vs_LYAM1_SYYWIGIR
51 и 120
0,016
0,676
IBP4_QCHPALDGQR_vs_IBP3_FLNVLSPR
2 и 99
0,028
0,661
B2MG_VNHVTLSQPK_vs_PGRP2_AGLLRPDYALLGHR
51 и 126
0,020
0,670
IBP4_QCHPALDGQR_vs_IBP3_YGQPLPGYTTK
2 и 100
0,011
0,685
B2MG_VNHVTLSQPK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
51 и 135
0,028
0,660
IBP4_QCHPALDGQR_vs_IGF2_GIVEECCFR
2 и 103
0,004
0,711
B2MG_VNHVTLSQPK_vs_SHBG_IALGGLLFPASNLR
51 и 18
0,019
0,671
IBP4_QCHPALDGQR_vs_ITIH4_ILDDLSPR
2 и 112
0,012
0,684
BGH3_LTLLAPLNSVFK_vs_CRIS3_AVSPPAR
52 и 78
0,033
0,656
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,005
0,705
BGH3_LTLLAPLNSVFK_vs_CRIS3_YEDLYSNCK
52 и 79
0,006
0,700
IBP4_QCHPALDGQR_vs_PGRP2_AGLLRPDYALLGHR
2 и 126
0,007
0,697
BGH3_LTLLAPLNSVFK_vs_IBP3_YGQPLPGYTTK
52 и 100
0,028
0,660
IBP4_QCHPALDGQR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
2 и 135
0,019
0,672
BGH3_LTLLAPLNSVFK_vs_IGF2_GIVEECCFR
52 и 103
0,010
0,688
IBP4_QCHPALDGQR_vs_PSG9_LFIPQITR
2 и 136
0,036
0,653
BGH3_LTLLAPLNSVFK_vs_LYAM1_SYYWIGIR
52 и 120
0,031
0,657
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,007
0,696
BGH3_LTLLAPLNSVFK_vs_PGRP2_AGLLRPDYALLGHR
52 и 126
0,036
0,653
IBP4_QCHPALDGQR_vs_SOM2.CSH_SVEGSCGF
2 и 139
0,018
0,673
BGH3_LTLLAPLNSVFK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
52 и 135
0,028
0,660
IBP4_QCHPALDGQR_vs_TENX_LNWEAPPGAFDSFLLR
2 и 141
0,025
0,664
BGH3_LTLLAPLNSVFK_vs_TENX_LNWEAPPGAFDSFLLR
52 и 141
0,047
0,645
IBP4_QCHPALDGQR_vs_VTDB_ELPEHTVK
2 и 147
0,038
0,651
C1QB_VPGLYYFTYHASSR_vs_IGF2_GIVEECCFR
55 и 103
0,041
0,649
INHBC_LDFHFSSDR_vs_ALS_IRPHTFTGLSGLR
107 и 40
0,028
0,660
C1QB_VPGLYYFTYHASSR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
55 и 135
0,040
0,650
INHBC_LDFHFSSDR_vs_CRIS3_AVSPPAR
107 и 78
0,026
0,663
CATD_VGFAEAAR_vs_ALS_IRPHTFTGLSGLR
57 и 40
0,022
0,667
INHBC_LDFHFSSDR_vs_CRIS3_YEDLYSNCK
107 и 79
0,012
0,684
CATD_VGFAEAAR_vs_CRIS3_AVSPPAR
57 и 78
0,015
0,678
INHBC_LDFHFSSDR_vs_IBP3_FLNVLSPR
107 и 99
0,011
0,685
CATD_VGFAEAAR_vs_CRIS3_YEDLYSNCK
57 и 79
0,006
0,701
INHBC_LDFHFSSDR_vs_IBP3_YGQPLPGYTTK
107 и 100
0,006
0,703
CATD_VGFAEAAR_vs_IBP3_FLNVLSPR
57 и 99
0,003
0,714
INHBC_LDFHFSSDR_vs_IGF2_GIVEECCFR
107 и 103
0,001
0,735
CATD_VGFAEAAR_vs_IBP3_YGQPLPGYTTK
57 и 100
0,002
0,726
INHBC_LDFHFSSDR_vs_ITIH4_ILDDLSPR
107 и 112
0,016
0,675
CATD_VGFAEAAR_vs_IGF2_GIVEECCFR
57 и 103
0,001
0,750
INHBC_LDFHFSSDR_vs_LYAM1_SYYWIGIR
107 и 120
0,016
0,675
CATD_VGFAEAAR_vs_ITIH4_ILDDLSPR
57 и 112
0,019
0,672
INHBC_LDFHFSSDR_vs_PGRP2_AGLLRPDYALLGHR
107 и 126
0,016
0,675
CATD_VGFAEAAR_vs_LYAM1_SYYWIGIR
57 и 120
0,014
0,679
INHBC_LDFHFSSDR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
107 и 135
0,019
0,671
CATD_VGFAEAAR_vs_NCAM1_GLGEISAASEFK
57 и 121
0,030
0,659
INHBC_LDFHFSSDR_vs_PSG9_LFIPQITR
107 и 136
0,033
0,656
CATD_VGFAEAAR_vs_PGRP2_AGLLRPDYALLGHR
57 и 126
0,008
0,693
INHBC_LDFHFSSDR_vs_SHBG_IALGGLLFPASNLR
107 и 18
0,013
0,681
CATD_VGFAEAAR_vs_PRG2_WNFAYWAAHQPWSR
57 и 129
0,041
0,649
INHBC_LDFHFSSDR_vs_SPRL1_VLTHSELAPLR
107 и 140
0,036
0,653
CATD_VGFAEAAR_vs_PSG3_VSAPSGTGHLPGLNPL
57 и 134
0,031
0,657
INHBC_LDFHFSSDR_vs_TENX_LNWEAPPGAFDSFLLR
107 и 141
0,023
0,666
CATD_VGFAEAAR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
57 и 135
0,011
0,686
INHBC_LDFHFSSDR_vs_VTDB_ELPEHTVK
107 и 147
0,025
0,664
CATD_VGFAEAAR_vs_PSG9_LFIPQITR
57 и 136
0,026
0,662
ITIH3_ALDLSLK_vs_CRIS3_AVSPPAR
111 и 78
0,040
0,650
CATD_VGFAEAAR_vs_SHBG_IALGGLLFPASNLR
57 и 18
0,026
0,663
ITIH3_ALDLSLK_vs_CRIS3_YEDLYSNCK
111 и 79
0,032
0,656
CATD_VGFAEAAR_vs_SOM2.CSH_SVEGSCGF
57 и 139
0,027
0,662
ITIH3_ALDLSLK_vs_IGF2_GIVEECCFR
111 и 103
0,044
0,647
CATD_VGFAEAAR_vs_SPRL1_VLTHSELAPLR
57 и 140
0,035
0,654
ITIH3_ALDLSLK_vs_LYAM1_SYYWIGIR
111 и 120
0,036
0,653
CATD_VGFAEAAR_vs_TENX_LNWEAPPGAFDSFLLR
57 и 141
0,005
0,707
ITIH3_ALDLSLK_vs_PGRP2_AGLLRPDYALLGHR
111 и 126
0,022
0,667
CATD_VGFAEAAR_vs_TENX_LSQLSVTDVTTSSLR
57 и 142
0,020
0,670
ITIH3_ALDLSLK_vs_SHBG_IALGGLLFPASNLR
111 и 18
0,017
0,675
CATD_VGFAEAAR_vs_VTDB_ELPEHTVK
57 и 147
0,026
0,662
KNG1_DIPTNSPELEETLTHTITK_vs_CRIS3_YEDLYSNCK
116 и 79
0,023
0,666
CATD_VSTLPAITLK_vs_ALS_IRPHTFTGLSGLR
58 и 40
0,013
0,681
KNG1_DIPTNSPELEETLTHTITK_vs_IGF2_GIVEECCFR
116 и 103
0,019
0,672
CATD_VSTLPAITLK_vs_CHL1_VIAVNEVGR
58 и 66
0,028
0,660
KNG1_DIPTNSPELEETLTHTITK_vs_LYAM1_SYYWIGIR
116 и 120
0,049
0,644
CATD_VSTLPAITLK_vs_CRIS3_AVSPPAR
58 и 78
0,009
0,692
KNG1_DIPTNSPELEETLTHTITK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
116 и 135
0,033
0,656
CATD_VSTLPAITLK_vs_CRIS3_YEDLYSNCK
58 и 79
0,004
0,712
KNG1_QVVAGLNFR_vs_CRIS3_AVSPPAR
117 и 78
0,044
0,647
CATD_VSTLPAITLK_vs_CSH_ISLLLIESWLEPVR
58 и 81
0,042
0,648
KNG1_QVVAGLNFR_vs_CRIS3_YEDLYSNCK
117 и 79
0,021
0,669
CATD_VSTLPAITLK_vs_IBP2_LIQGAPTIR
58 и 98
0,038
0,651
KNG1_QVVAGLNFR_vs_IBP3_FLNVLSPR
117 и 99
0,028
0,661
CATD_VSTLPAITLK_vs_IBP3_FLNVLSPR
58 и 99
0,002
0,730
KNG1_QVVAGLNFR_vs_IBP3_YGQPLPGYTTK
117 и 100
0,014
0,680
CATD_VSTLPAITLK_vs_IBP3_YGQPLPGYTTK
58 и 100
0,001
0,737
KNG1_QVVAGLNFR_vs_IGF2_GIVEECCFR
117 и 103
0,007
0,697
CATD_VSTLPAITLK_vs_IGF2_GIVEECCFR
58 и 103
0,000
0,762
KNG1_QVVAGLNFR_vs_ITIH4_ILDDLSPR
117 и 112
0,039
0,651
CATD_VSTLPAITLK_vs_ITIH4_ILDDLSPR
58 и 112
0,007
0,695
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,017
0,674
CATD_VSTLPAITLK_vs_LYAM1_SYYWIGIR
58 и 120
0,010
0,687
KNG1_QVVAGLNFR_vs_PGRP2_AGLLRPDYALLGHR
117 и 126
0,034
0,655
CATD_VSTLPAITLK_vs_NCAM1_GLGEISAASEFK
58 и 121
0,024
0,665
KNG1_QVVAGLNFR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
117 и 135
0,028
0,660
CATD_VSTLPAITLK_vs_PGRP2_AGLLRPDYALLGHR
58 и 126
0,002
0,727
KNG1_QVVAGLNFR_vs_SHBG_IALGGLLFPASNLR
117 и 18
0,031
0,657
CATD_VSTLPAITLK_vs_PSG3_VSAPSGTGHLPGLNPL
58 и 134
0,038
0,651
LBP_ITGFLKPGK_vs_CRIS3_AVSPPAR
118 и 78
0,018
0,673
CATD_VSTLPAITLK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
58 и 135
0,012
0,684
LBP_ITGFLKPGK_vs_CRIS3_YEDLYSNCK
118 и 79
0,006
0,700
CATD_VSTLPAITLK_vs_PSG9_LFIPQITR
58 и 136
0,019
0,672
LBP_ITGFLKPGK_vs_IBP3_FLNVLSPR
118 и 99
0,039
0,651
CATD_VSTLPAITLK_vs_SHBG_IALGGLLFPASNLR
58 и 18
0,007
0,695
LBP_ITGFLKPGK_vs_IBP3_YGQPLPGYTTK
118 и 100
0,017
0,674
CATD_VSTLPAITLK_vs_SOM2.CSH_SVEGSCGF
58 и 139
0,027
0,662
LBP_ITGFLKPGK_vs_IGF2_GIVEECCFR
118 и 103
0,006
0,700
CATD_VSTLPAITLK_vs_SPRL1_VLTHSELAPLR
58 и 140
0,029
0,659
LBP_ITGFLKPGK_vs_ITIH4_ILDDLSPR
118 и 112
0,037
0,652
CATD_VSTLPAITLK_vs_TENX_LNWEAPPGAFDSFLLR
58 и 141
0,002
0,722
LBP_ITGFLKPGK_vs_LYAM1_SYYWIGIR
118 и 120
0,016
0,676
CATD_VSTLPAITLK_vs_TENX_LSQLSVTDVTTSSLR
58 и 142
0,007
0,697
LBP_ITGFLKPGK_vs_PGRP2_AGLLRPDYALLGHR
118 и 126
0,023
0,666
CATD_VSTLPAITLK_vs_VTDB_ELPEHTVK
58 и 147
0,029
0,659
LBP_ITGFLKPGK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
118 и 135
0,013
0,682
CBPN_EALIQFLEQVHQGIK_vs_IBP3_YGQPLPGYTTK
59 и 100
0,037
0,652
LBP_ITGFLKPGK_vs_PSG9_LFIPQITR
118 и 136
0,020
0,670
CBPN_EALIQFLEQVHQGIK_vs_IGF2_GIVEECCFR
59 и 103
0,028
0,660
LBP_ITGFLKPGK_vs_SHBG_IALGGLLFPASNLR
118 и 18
0,014
0,680
CBPN_NNANGVDLNR_vs_CRIS3_YEDLYSNCK
60 и 79
0,043
0,648
LBP_ITGFLKPGK_vs_SOM2.CSH_SVEGSCGF
118 и 139
0,024
0,664
CBPN_NNANGVDLNR_vs_IBP3_FLNVLSPR
60 и 99
0,032
0,656
LBP_ITGFLKPGK_vs_SPRL1_VLTHSELAPLR
118 и 140
0,044
0,647
CBPN_NNANGVDLNR_vs_IBP3_YGQPLPGYTTK
60 и 100
0,013
0,681
LBP_ITGFLKPGK_vs_VTDB_ELPEHTVK
118 и 147
0,031
0,658
CBPN_NNANGVDLNR_vs_IGF2_GIVEECCFR
60 и 103
0,006
0,701
LBP_ITLPDFTGDLR_vs_ALS_IRPHTFTGLSGLR
119 и 40
0,031
0,657
CBPN_NNANGVDLNR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
60 и 135
0,044
0,647
LBP_ITLPDFTGDLR_vs_CRIS3_AVSPPAR
119 и 78
0,004
0,710
CBPN_NNANGVDLNR_vs_SHBG_IALGGLLFPASNLR
60 и 18
0,038
0,651
LBP_ITLPDFTGDLR_vs_CRIS3_YEDLYSNCK
119 и 79
0,002
0,727
CD14_LTVGAAQVPAQLLVGALR_vs_IGF2_GIVEECCFR
61 и 103
0,033
0,656
LBP_ITLPDFTGDLR_vs_IBP2_LIQGAPTIR
119 и 98
0,031
0,658
CFAB_YGLVTYATYPK_vs_IBP3_FLNVLSPR
64 и 99
0,032
0,656
LBP_ITLPDFTGDLR_vs_IBP3_FLNVLSPR
119 и 99
0,007
0,698
CFAB_YGLVTYATYPK_vs_IBP3_YGQPLPGYTTK
64 и 100
0,013
0,681
LBP_ITLPDFTGDLR_vs_IBP3_YGQPLPGYTTK
119 и 100
0,003
0,717
CFAB_YGLVTYATYPK_vs_IGF2_GIVEECCFR
64 и 103
0,010
0,689
LBP_ITLPDFTGDLR_vs_IGF2_GIVEECCFR
119 и 103
0,001
0,733
CFAB_YGLVTYATYPK_vs_LYAM1_SYYWIGIR
64 и 120
0,035
0,654
LBP_ITLPDFTGDLR_vs_ITIH4_ILDDLSPR
119 и 112
0,004
0,710
CFAB_YGLVTYATYPK_vs_PGRP2_AGLLRPDYALLGHR
64 и 126
0,048
0,645
LBP_ITLPDFTGDLR_vs_LYAM1_SYYWIGIR
119 и 120
0,003
0,716
CLUS_ASSIIDELFQDR_vs_IBP3_YGQPLPGYTTK
67 и 100
0,049
0,644
LBP_ITLPDFTGDLR_vs_NCAM1_GLGEISAASEFK
119 и 121
0,045
0,646
CLUS_ASSIIDELFQDR_vs_IGF2_GIVEECCFR
67 и 103
0,027
0,662
LBP_ITLPDFTGDLR_vs_PGRP2_AGLLRPDYALLGHR
119 и 126
0,002
0,722
CO8A_SLLQPNK_vs_IGF2_GIVEECCFR
74 и 103
0,030
0,659
LBP_ITLPDFTGDLR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
119 и 135
0,005
0,703
CO8B_QALEEFQK_vs_IBP3_YGQPLPGYTTK
76 и 100
0,039
0,651
LBP_ITLPDFTGDLR_vs_PSG9_LFIPQITR
119 и 136
0,010
0,689
CO8B_QALEEFQK_vs_IGF2_GIVEECCFR
76 и 103
0,017
0,675
LBP_ITLPDFTGDLR_vs_SHBG_IALGGLLFPASNLR
119 и 18
0,003
0,716
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_ALS_IRPHTFTGLSGLR
82 и 40
0,001
0,737
LBP_ITLPDFTGDLR_vs_SOM2.CSH_SVEGSCGF
119 и 139
0,011
0,686
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_C163A_INPASLDK
82 и 54
0,019
0,672
LBP_ITLPDFTGDLR_vs_SPRL1_VLTHSELAPLR
119 и 140
0,013
0,681
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_CHL1_VIAVNEVGR
82 и 66
0,013
0,681
LBP_ITLPDFTGDLR_vs_TENX_LNWEAPPGAFDSFLLR
119 и 141
0,014
0,680
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_CRIS3_AVSPPAR
82 и 78
0,006
0,700
LBP_ITLPDFTGDLR_vs_TENX_LSQLSVTDVTTSSLR
119 и 142
0,044
0,647
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_CRIS3_YEDLYSNCK
82 и 79
0,003
0,716
LBP_ITLPDFTGDLR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
119 и 144
0,039
0,651
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_CSH_AHQLAIDTYQEFEETYIPK
82 и 80
0,011
0,686
LBP_ITLPDFTGDLR_vs_VTDB_ELPEHTVK
119 и 147
0,006
0,701
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_CSH_ISLLLIESWLEPVR
82 и 81
0,012
0,684
PEDF_LQSLFDSPDFSK_vs_CRIS3_AVSPPAR
124 и 78
0,022
0,667
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_FBLN1_TGYYFDGISR
82 и 86
0,037
0,653
PEDF_LQSLFDSPDFSK_vs_CRIS3_YEDLYSNCK
124 и 79
0,009
0,690
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_IBP2_LIQGAPTIR
82 и 98
0,008
0,692
PEDF_LQSLFDSPDFSK_vs_IBP3_YGQPLPGYTTK
124 и 100
0,045
0,646
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_IBP3_FLNVLSPR
82 и 99
0,001
0,744
PEDF_LQSLFDSPDFSK_vs_IGF2_GIVEECCFR
124 и 103
0,011
0,686
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_IBP3_YGQPLPGYTTK
82 и 100
0,000
0,758
PEDF_LQSLFDSPDFSK_vs_ITIH4_ILDDLSPR
124 и 112
0,049
0,644
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_IGF2_GIVEECCFR
82 и 103
0,000
0,767
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,024
0,665
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_ITIH4_ILDDLSPR
82 и 112
0,001
0,748
PEDF_LQSLFDSPDFSK_vs_PGRP2_AGLLRPDYALLGHR
124 и 126
0,035
0,654
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_LYAM1_SYYWIGIR
82 и 120
0,001
0,748
PEDF_LQSLFDSPDFSK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
124 и 135
0,037
0,653
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_NCAM1_GLGEISAASEFK
82 и 121
0,010
0,687
PEDF_TVQAVLTVPK_vs_CRIS3_YEDLYSNCK
125 и 79
0,037
0,652
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PGRP2_AGLLRPDYALLGHR
82 и 126
0,001
0,742
PTGDS_GPGEDFR_vs_CRIS3_YEDLYSNCK
137 и 79
0,041
0,649
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PRG2_WNFAYWAAHQPWSR
82 и 129
0,033
0,656
PTGDS_GPGEDFR_vs_IGF2_GIVEECCFR
137 и 103
0,024
0,665
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PSG3_VSAPSGTGHLPGLNPL
82 и 134
0,015
0,677
THBG_AVLHIGEK_vs_CRIS3_YEDLYSNCK
143 и 79
0,050
0,643
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
82 и 135
0,002
0,726
VTNC_GQYCYELDEK_vs_CRIS3_AVSPPAR
149 и 78
0,047
0,645
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PSG9_LFIPQITR
82 и 136
0,005
0,703
VTNC_GQYCYELDEK_vs_CRIS3_YEDLYSNCK
149 и 79
0,024
0,665
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_SHBG_IALGGLLFPASNLR
82 и 18
0,001
0,747
VTNC_GQYCYELDEK_vs_IBP3_FLNVLSPR
149 и 99
0,011
0,686
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_SOM2.CSH_NYGLLYCFR
82 и 138
0,030
0,659
VTNC_GQYCYELDEK_vs_IBP3_YGQPLPGYTTK
149 и 100
0,003
0,718
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_SOM2.CSH_SVEGSCGF
82 и 139
0,005
0,707
VTNC_GQYCYELDEK_vs_IGF2_GIVEECCFR
149 и 103
0,003
0,719
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_SPRL1_VLTHSELAPLR
82 и 140
0,007
0,698
VTNC_GQYCYELDEK_vs_ITIH4_ILDDLSPR
149 и 112
0,017
0,675
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_TENX_LNWEAPPGAFDSFLLR
82 и 141
0,005
0,706
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,026
0,663
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_TENX_LSQLSVTDVTTSSLR
82 и 142
0,013
0,681
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,042
0,648
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
82 и 144
0,009
0,691
VTNC_GQYCYELDEK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
149 и 135
0,028
0,660
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_VTDB_ELPEHTVK
82 и 147
0,002
0,722
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,013
0,681
ENPP2_TYLHTYESEI_vs_ALS_IRPHTFTGLSGLR
83 и 40
0,001
0,733
VTNC_GQYCYELDEK_vs_VTDB_ELPEHTVK
149 и 147
0,037
0,653
ENPP2_TYLHTYESEI_vs_C163A_INPASLDK
83 и 54
0,015
0,678
VTNC_VDTVDPPYPR_vs_CRIS3_AVSPPAR
150 и 78
0,037
0,652
ENPP2_TYLHTYESEI_vs_CHL1_VIAVNEVGR
83 и 66
0,018
0,673
VTNC_VDTVDPPYPR_vs_CRIS3_YEDLYSNCK
150 и 79
0,022
0,667
ENPP2_TYLHTYESEI_vs_CRIS3_AVSPPAR
83 и 78
0,009
0,692
VTNC_VDTVDPPYPR_vs_IBP3_FLNVLSPR
150 и 99
0,015
0,677
ENPP2_TYLHTYESEI_vs_CRIS3_YEDLYSNCK
83 и 79
0,003
0,716
VTNC_VDTVDPPYPR_vs_IBP3_YGQPLPGYTTK
150 и 100
0,006
0,699
ENPP2_TYLHTYESEI_vs_CSH_AHQLAIDTYQEFEETYIPK
83 и 80
0,007
0,695
VTNC_VDTVDPPYPR_vs_IGF2_GIVEECCFR
150 и 103
0,004
0,710
ENPP2_TYLHTYESEI_vs_CSH_ISLLLIESWLEPVR
83 и 81
0,010
0,689
VTNC_VDTVDPPYPR_vs_ITIH4_ILDDLSPR
150 и 112
0,037
0,653
ENPP2_TYLHTYESEI_vs_IBP2_LIQGAPTIR
83 и 98
0,010
0,689
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,018
0,673
ENPP2_TYLHTYESEI_vs_IBP3_FLNVLSPR
83 и 99
0,002
0,728
VTNC_VDTVDPPYPR_vs_PGRP2_AGLLRPDYALLGHR
150 и 126
0,044
0,647
ENPP2_TYLHTYESEI_vs_IBP3_YGQPLPGYTTK
83 и 100
0,000
0,755
VTNC_VDTVDPPYPR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
150 и 135
0,021
0,668
ENPP2_TYLHTYESEI_vs_IGF2_GIVEECCFR
83 и 103
0,001
0,749
VTNC_VDTVDPPYPR_vs_PSG9_LFIPQITR
150 и 136
0,041
0,649
ENPP2_TYLHTYESEI_vs_ITIH4_ILDDLSPR
83 и 112
0,002
0,727
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,012
0,684
ENPP2_TYLHTYESEI_vs_LYAM1_SYYWIGIR
83 и 120
0,001
0,733
VTNC_VDTVDPPYPR_vs_VTDB_ELPEHTVK
150 и 147
0,028
0,660
ENPP2_TYLHTYESEI_vs_NCAM1_GLGEISAASEFK
83 и 121
0,020
0,670
83 и 126
Таблица 41: эффективность классификации по обращениям, недели 20 и 21.
AUROC обращения для недель беременности 20 и 21 с использованием порогов случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, без BMI-стратификации.
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
AFAM_DADPDTFFAK_vs_IBP2_LIQGAPTIR
37 и 98
0,042
0,703
CATD_VSTLPAITLK_vs_PSG9_LFIPQITR
58 и 136
0,033
0,714
AFAM_DADPDTFFAK_vs_PGRP2_AGLLRPDYALLGHR
37 и 126
0,045
0,701
CATD_VSTLPAITLK_vs_SHBG_IALGGLLFPASNLR
58 и 18
0,002
0,803
AFAM_DADPDTFFAK_vs_SHBG_IALGGLLFPASNLR
37 и 18
0,036
0,709
CATD_VSTLPAITLK_vs_SOM2.CSH_NYGLLYCFR
58 и 138
0,003
0,796
AFAM_HFQNLGK_vs_ALS_IRPHTFTGLSGLR
38 и 40
0,020
0,732
CATD_VSTLPAITLK_vs_SOM2.CSH_SVEGSCGF
58 и 139
0,003
0,796
AFAM_HFQNLGK_vs_IBP2_LIQGAPTIR
38 и 98
0,024
0,726
CATD_VSTLPAITLK_vs_SPRL1_VLTHSELAPLR
58 и 140
0,001
0,830
AFAM_HFQNLGK_vs_IBP3_YGQPLPGYTTK
38 и 100
0,048
0,698
CATD_VSTLPAITLK_vs_TENX_LNWEAPPGAFDSFLLR
58 и 141
0,001
0,820
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,048
0,698
CATD_VSTLPAITLK_vs_TENX_LSQLSVTDVTTSSLR
58 и 142
0,008
0,767
AFAM_HFQNLGK_vs_ITIH4_ILDDLSPR
38 и 112
0,033
0,714
CATD_VSTLPAITLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
58 и 144
0,009
0,760
AFAM_HFQNLGK_vs_LYAM1_SYYWIGIR
38 и 120
0,031
0,715
CATD_VSTLPAITLK_vs_VTDB_ELPEHTVK
58 и 147
0,001
0,819
AFAM_HFQNLGK_vs_PGRP2_AGLLRPDYALLGHR
38 и 126
0,017
0,738
CBPN_EALIQFLEQVHQGIK_vs_SHBG_IALGGLLFPASNLR
59 и 18
0,008
0,764
AFAM_HFQNLGK_vs_PSG3_VSAPSGTGHLPGLNPL
38 и 134
0,029
0,719
CBPN_NNANGVDLNR_vs_SHBG_IALGGLLFPASNLR
60 и 18
0,016
0,740
AFAM_HFQNLGK_vs_SHBG_IALGGLLFPASNLR
38 и 18
0,017
0,738
CBPN_NNANGVDLNR_vs_SPRL1_VLTHSELAPLR
60 и 140
0,037
0,709
AFAM_HFQNLGK_vs_SOM2.CSH_SVEGSCGF
38 и 139
0,048
0,698
CD14_LTVGAAQVPAQLLVGALR_vs_PGRP2_AGLLRPDYALLGHR
61 и 126
0,034
0,712
AFAM_HFQNLGK_vs_SPRL1_VLTHSELAPLR
38 и 140
0,018
0,737
CD14_LTVGAAQVPAQLLVGALR_vs_SHBG_IALGGLLFPASNLR
61 и 18
0,027
0,721
AFAM_HFQNLGK_vs_VTDB_ELPEHTVK
38 и 147
0,014
0,746
CD14_SWLAELQQWLKPGLK_vs_LYAM1_SYYWIGIR
62 и 120
0,038
0,708
ANGT_DPTFIPAPIQAK_vs_SHBG_IALGGLLFPASNLR
42 и 18
0,019
0,735
CD14_SWLAELQQWLKPGLK_vs_PGRP2_AGLLRPDYALLGHR
62 и 126
0,029
0,718
APOH_ATVVYQGER_vs_SHBG_IALGGLLFPASNLR
48 и 18
0,045
0,701
CD14_SWLAELQQWLKPGLK_vs_SHBG_IALGGLLFPASNLR
62 и 18
0,026
0,722
B2MG_VEHSDLSFSK_vs_IBP2_LIQGAPTIR
50 и 98
0,049
0,697
CD14_SWLAELQQWLKPGLK_vs_VTDB_ELPEHTVK
62 и 147
0,021
0,731
B2MG_VEHSDLSFSK_vs_SHBG_IALGGLLFPASNLR
50 и 18
0,029
0,718
CO5_TLLPVSKPEIR_vs_SHBG_IALGGLLFPASNLR
70 и 18
0,045
0,701
B2MG_VNHVTLSQPK_vs_IBP2_LIQGAPTIR
51 и 98
0,049
0,697
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PGRP2_AGLLRPDYALLGHR
82 и 126
0,045
0,701
B2MG_VNHVTLSQPK_vs_LYAM1_SYYWIGIR
51 и 120
0,031
0,716
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_SHBG_IALGGLLFPASNLR
82 и 18
0,032
0,715
B2MG_VNHVTLSQPK_vs_PGRP2_AGLLRPDYALLGHR
51 и 126
0,035
0,711
ENPP2_TYLHTYESEI_vs_ALS_IRPHTFTGLSGLR
83 и 40
0,031
0,715
B2MG_VNHVTLSQPK_vs_SHBG_IALGGLLFPASNLR
51 и 18
0,028
0,720
ENPP2_TYLHTYESEI_vs_IBP1_VVESLAK
83 и 97
0,047
0,699
B2MG_VNHVTLSQPK_vs_SOM2.CSH_SVEGSCGF
51 и 139
0,049
0,697
ENPP2_TYLHTYESEI_vs_IBP2_LIQGAPTIR
83 и 98
0,038
0,708
B2MG_VNHVTLSQPK_vs_SPRL1_VLTHSELAPLR
51 и 140
0,046
0,700
ENPP2_TYLHTYESEI_vs_ITIH4_ILDDLSPR
83 и 112
0,047
0,699
CATD_VGFAEAAR_vs_ALS_IRPHTFTGLSGLR
57 и 40
0,001
0,828
ENPP2_TYLHTYESEI_vs_LYAM1_SYYWIGIR
83 и 120
0,026
0,722
CATD_VGFAEAAR_vs_C163A_INPASLDK
57 и 54
0,001
0,832
ENPP2_TYLHTYESEI_vs_PGRP2_AGLLRPDYALLGHR
83 и 126
0,020
0,732
CATD_VGFAEAAR_vs_CHL1_VIAVNEVGR
57 и 66
0,023
0,728
ENPP2_TYLHTYESEI_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
83 и 135
0,049
0,697
CATD_VGFAEAAR_vs_CRIS3_AVSPPAR
57 и 78
0,017
0,738
ENPP2_TYLHTYESEI_vs_SHBG_IALGGLLFPASNLR
83 и 18
0,017
0,739
CATD_VGFAEAAR_vs_CRIS3_YEDLYSNCK
57 и 79
0,018
0,737
ENPP2_TYLHTYESEI_vs_SOM2.CSH_SVEGSCGF
83 и 139
0,036
0,710
CATD_VGFAEAAR_vs_CSH_AHQLAIDTYQEFEETYIPK
57 и 80
0,001
0,824
ENPP2_TYLHTYESEI_vs_SPRL1_VLTHSELAPLR
83 и 140
0,028
0,720
CATD_VGFAEAAR_vs_CSH_ISLLLIESWLEPVR
57 и 81
0,001
0,819
ENPP2_TYLHTYESEI_vs_VTDB_ELPEHTVK
83 и 147
0,025
0,725
CATD_VGFAEAAR_vs_FBLN1_TGYYFDGISR
57 и 86
0,001
0,832
F13B_GDTYPAELYITGSILR_vs_IBP2_LIQGAPTIR
84 и 98
0,031
0,715
CATD_VGFAEAAR_vs_IBP1_VVESLAK
57 и 97
0,004
0,791
F13B_GDTYPAELYITGSILR_vs_LYAM1_SYYWIGIR
84 и 120
0,042
0,703
CATD_VGFAEAAR_vs_IBP2_LIQGAPTIR
57 и 98
0,003
0,793
F13B_GDTYPAELYITGSILR_vs_PGRP2_AGLLRPDYALLGHR
84 и 126
0,040
0,706
CATD_VGFAEAAR_vs_IBP3_FLNVLSPR
57 и 99
0,012
0,751
F13B_GDTYPAELYITGSILR_vs_SHBG_IALGGLLFPASNLR
84 и 18
0,021
0,731
CATD_VGFAEAAR_vs_IBP3_YGQPLPGYTTK
57 и 100
0,007
0,768
F13B_GDTYPAELYITGSILR_vs_SPRL1_VLTHSELAPLR
84 и 140
0,042
0,703
CATD_VGFAEAAR_vs_IGF2_GIVEECCFR
57 и 103
0,006
0,776
FETUA_FSVVYAK_vs_IBP2_LIQGAPTIR
88 и 98
0,026
0,722
CATD_VGFAEAAR_vs_ITIH4_ILDDLSPR
57 и 112
0,003
0,797
FETUA_FSVVYAK_vs_ITIH4_ILDDLSPR
88 и 112
0,024
0,726
CATD_VGFAEAAR_vs_LYAM1_SYYWIGIR
57 и 120
0,001
0,832
FETUA_FSVVYAK_vs_LYAM1_SYYWIGIR
88 и 120
0,010
0,756
CATD_VGFAEAAR_vs_NCAM1_GLGEISAASEFK
57 и 121
0,010
0,756
FETUA_FSVVYAK_vs_PGRP2_AGLLRPDYALLGHR
88 и 126
0,031
0,715
CATD_VGFAEAAR_vs_PGRP2_AGLLRPDYALLGHR
57 и 126
0,000
0,859
FETUA_FSVVYAK_vs_PSG3_VSAPSGTGHLPGLNPL
88 и 134
0,043
0,703
CATD_VGFAEAAR_vs_PRG2_WNFAYWAAHQPWSR
57 и 129
0,001
0,822
FETUA_FSVVYAK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
88 и 135
0,045
0,701
CATD_VGFAEAAR_vs_PSG1_FQLPGQK
57 и 131
0,009
0,760
FETUA_FSVVYAK_vs_SHBG_IALGGLLFPASNLR
88 и 18
0,021
0,731
CATD_VGFAEAAR_vs_PSG3_VSAPSGTGHLPGLNPL
57 и 134
0,001
0,821
FETUA_FSVVYAK_vs_SOM2.CSH_SVEGSCGF
88 и 139
0,031
0,716
CATD_VGFAEAAR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
57 и 135
0,006
0,777
FETUA_FSVVYAK_vs_SPRL1_VLTHSELAPLR
88 и 140
0,016
0,741
CATD_VGFAEAAR_vs_PSG9_LFIPQITR
57 и 136
0,014
0,747
FETUA_FSVVYAK_vs_VTDB_ELPEHTVK
88 и 147
0,001
0,818
CATD_VGFAEAAR_vs_SHBG_IALGGLLFPASNLR
57 и 18
0,003
0,802
FETUA_HTLNQIDEVK_vs_ALS_IRPHTFTGLSGLR
89 и 40
0,033
0,713
CATD_VGFAEAAR_vs_SOM2.CSH_NYGLLYCFR
57 и 138
0,001
0,823
FETUA_HTLNQIDEVK_vs_IBP2_LIQGAPTIR
89 и 98
0,018
0,737
CATD_VGFAEAAR_vs_SOM2.CSH_SVEGSCGF
57 и 139
0,001
0,831
FETUA_HTLNQIDEVK_vs_ITIH4_ILDDLSPR
89 и 112
0,022
0,729
CATD_VGFAEAAR_vs_SPRL1_VLTHSELAPLR
57 и 140
0,000
0,864
FETUA_HTLNQIDEVK_vs_LYAM1_SYYWIGIR
89 и 120
0,012
0,751
CATD_VGFAEAAR_vs_TENX_LNWEAPPGAFDSFLLR
57 и 141
0,001
0,819
FETUA_HTLNQIDEVK_vs_PGRP2_AGLLRPDYALLGHR
89 и 126
0,028
0,720
CATD_VGFAEAAR_vs_TENX_LSQLSVTDVTTSSLR
57 и 142
0,006
0,776
FETUA_HTLNQIDEVK_vs_PSG3_VSAPSGTGHLPGLNPL
89 и 134
0,024
0,726
CATD_VGFAEAAR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
57 и 144
0,005
0,779
FETUA_HTLNQIDEVK_vs_SHBG_IALGGLLFPASNLR
89 и 18
0,007
0,768
CATD_VGFAEAAR_vs_VTDB_ELPEHTVK
57 и 147
0,000
0,855
FETUA_HTLNQIDEVK_vs_SPRL1_VLTHSELAPLR
89 и 140
0,008
0,764
CATD_VSTLPAITLK_vs_ALS_IRPHTFTGLSGLR
58 и 40
0,001
0,826
FETUA_HTLNQIDEVK_vs_VTDB_ELPEHTVK
89 и 147
0,003
0,792
CATD_VSTLPAITLK_vs_C163A_INPASLDK
58 и 54
0,012
0,752
HEMO_NFPSPVDAAFR_vs_SHBG_IALGGLLFPASNLR
93 и 18
0,038
0,708
CATD_VSTLPAITLK_vs_CHL1_VIAVNEVGR
58 и 66
0,023
0,727
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,036
0,709
CATD_VSTLPAITLK_vs_CRIS3_AVSPPAR
58 и 78
0,029
0,718
IBP6_GAQTLYVPNCDHR_vs_TENX_LSQLSVTDVTTSSLR
101 и 142
0,044
0,702
CATD_VSTLPAITLK_vs_CRIS3_YEDLYSNCK
58 и 79
0,028
0,720
ITIH3_ALDLSLK_vs_SHBG_IALGGLLFPASNLR
111 и 18
0,021
0,732
CATD_VSTLPAITLK_vs_CSH_AHQLAIDTYQEFEETYIPK
58 и 80
0,003
0,798
ITIH4_NPLVWVHASPEHVVVTR_vs_ITIH4_ILDDLSPR
113 и 112
0,022
0,729
CATD_VSTLPAITLK_vs_CSH_ISLLLIESWLEPVR
58 и 81
0,003
0,800
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,040
0,706
CATD_VSTLPAITLK_vs_FBLN1_TGYYFDGISR
58 и 86
0,001
0,838
KNG1_QVVAGLNFR_vs_PGRP2_AGLLRPDYALLGHR
117 и 126
0,025
0,725
CATD_VSTLPAITLK_vs_IBP1_VVESLAK
58 и 97
0,004
0,791
KNG1_QVVAGLNFR_vs_SHBG_IALGGLLFPASNLR
117 и 18
0,006
0,773
CATD_VSTLPAITLK_vs_IBP2_LIQGAPTIR
58 и 98
0,003
0,797
KNG1_QVVAGLNFR_vs_VTDB_ELPEHTVK
117 и 147
0,049
0,697
CATD_VSTLPAITLK_vs_IBP3_FLNVLSPR
58 и 99
0,006
0,774
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,033
0,713
CATD_VSTLPAITLK_vs_IBP3_YGQPLPGYTTK
58 и 100
0,006
0,774
PEDF_LQSLFDSPDFSK_vs_SHBG_IALGGLLFPASNLR
124 и 18
0,035
0,711
CATD_VSTLPAITLK_vs_IGF2_GIVEECCFR
58 и 103
0,005
0,779
PEDF_LQSLFDSPDFSK_vs_VTDB_ELPEHTVK
124 и 147
0,034
0,712
CATD_VSTLPAITLK_vs_ITIH4_ILDDLSPR
58 и 112
0,008
0,767
PRDX2_GLFIIDGK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
128 и 135
0,049
0,697
CATD_VSTLPAITLK_vs_LYAM1_SYYWIGIR
58 и 120
0,002
0,805
PRDX2_GLFIIDGK_vs_SHBG_IALGGLLFPASNLR
128 и 18
0,035
0,711
CATD_VSTLPAITLK_vs_NCAM1_GLGEISAASEFK
58 и 121
0,012
0,750
PTGDS_GPGEDFR_vs_SHBG_IALGGLLFPASNLR
137 и 18
0,041
0,704
CATD_VSTLPAITLK_vs_PGRP2_AGLLRPDYALLGHR
58 и 126
0,001
0,844
THBG_AVLHIGEK_vs_VTDB_ELPEHTVK
143 и 147
0,023
0,728
CATD_VSTLPAITLK_vs_PRG2_WNFAYWAAHQPWSR
58 и 129
0,004
0,785
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,026
0,723
CATD_VSTLPAITLK_vs_PSG1_FQLPGQK
58 и 131
0,016
0,740
VTNC_GQYCYELDEK_vs_VTDB_ELPEHTVK
149 и 147
0,032
0,715
CATD_VSTLPAITLK_vs_PSG3_VSAPSGTGHLPGLNPL
58 и 134
0,001
0,831
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,033
0,714
CATD_VSTLPAITLK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
58 и 135
0,018
0,736
Таблица 42: эффективность классификации по обращениям, недели 20 и 21.
AUROC обращения для недель беременности 20 и 21 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, с BMI-стратификацией (>22≤37).
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
A2GL_DLLLPQPDLR_vs_IBP1_VVESLAK
34 и 97
0,042
0,734
CD14_SWLAELQQWLKPGLK_vs_SHBG_IALGGLLFPASNLR
62 и 18
0,024
0,759
A2GL_DLLLPQPDLR_vs_IBP2_LIQGAPTIR
34 и 98
0,043
0,732
CD14_SWLAELQQWLKPGLK_vs_TENX_LSQLSVTDVTTSSLR
62 и 142
0,036
0,741
A2GL_DLLLPQPDLR_vs_SHBG_IALGGLLFPASNLR
34 и 18
0,033
0,745
CFAB_YGLVTYATYPK_vs_SHBG_IALGGLLFPASNLR
64 и 18
0,042
0,734
A2GL_DLLLPQPDLR_vs_SOM2.CSH_SVEGSCGF
34 и 139
0,048
0,727
CLUS_ASSIIDELFQDR_vs_SHBG_IALGGLLFPASNLR
67 и 18
0,037
0,740
AFAM_DADPDTFFAK_vs_IBP2_LIQGAPTIR
37 и 98
0,034
0,743
CLUS_ASSIIDELFQDR_vs_TENX_LSQLSVTDVTTSSLR
67 и 142
0,018
0,772
AFAM_DADPDTFFAK_vs_ITIH4_ILDDLSPR
37 и 112
0,032
0,747
CLUS_LFDSDPITVTVPVEVSR_vs_TENX_LSQLSVTDVTTSSLR
68 и 142
0,008
0,803
AFAM_DADPDTFFAK_vs_LYAM1_SYYWIGIR
37 и 120
0,023
0,761
CO5_TLLPVSKPEIR_vs_SHBG_IALGGLLFPASNLR
70 и 18
0,045
0,731
AFAM_DADPDTFFAK_vs_PGRP2_AGLLRPDYALLGHR
37 и 126
0,032
0,747
CO5_TLLPVSKPEIR_vs_TENX_LSQLSVTDVTTSSLR
70 и 142
0,012
0,790
AFAM_DADPDTFFAK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
37 и 135
0,043
0,732
CO5_VFQFLEK_vs_TENX_LSQLSVTDVTTSSLR
71 и 142
0,026
0,756
AFAM_DADPDTFFAK_vs_SHBG_IALGGLLFPASNLR
37 и 18
0,015
0,779
CO6_ALNHLPLEYNSALYSR_vs_SHBG_IALGGLLFPASNLR
72 и 18
0,029
0,750
AFAM_DADPDTFFAK_vs_SOM2.CSH_SVEGSCGF
37 и 139
0,039
0,738
CO6_ALNHLPLEYNSALYSR_vs_TENX_LSQLSVTDVTTSSLR
72 и 142
0,007
0,810
AFAM_HFQNLGK_vs_ALS_IRPHTFTGLSGLR
38 и 40
0,019
0,769
CO8B_QALEEFQK_vs_IBP1_VVESLAK
76 и 97
0,043
0,732
AFAM_HFQNLGK_vs_IBP1_VVESLAK
38 и 97
0,042
0,734
CO8B_QALEEFQK_vs_SHBG_IALGGLLFPASNLR
76 и 18
0,033
0,745
AFAM_HFQNLGK_vs_IBP2_LIQGAPTIR
38 и 98
0,014
0,781
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_IBP1_VVESLAK
82 и 97
0,048
0,727
AFAM_HFQNLGK_vs_ITIH4_ILDDLSPR
38 и 112
0,008
0,807
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_IBP2_LIQGAPTIR
82 и 98
0,040
0,736
AFAM_HFQNLGK_vs_LYAM1_SYYWIGIR
38 и 120
0,006
0,817
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PGRP2_AGLLRPDYALLGHR
82 и 126
0,043
0,732
AFAM_HFQNLGK_vs_PGRP2_AGLLRPDYALLGHR
38 и 126
0,010
0,797
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
82 и 135
0,033
0,745
AFAM_HFQNLGK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
38 и 135
0,033
0,745
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_SHBG_IALGGLLFPASNLR
82 и 18
0,032
0,747
AFAM_HFQNLGK_vs_SHBG_IALGGLLFPASNLR
38 и 18
0,006
0,816
ENPP2_TYLHTYESEI_vs_IBP1_VVESLAK
83 и 97
0,045
0,731
AFAM_HFQNLGK_vs_SOM2.CSH_SVEGSCGF
38 и 139
0,014
0,783
ENPP2_TYLHTYESEI_vs_IBP2_LIQGAPTIR
83 и 98
0,039
0,738
AFAM_HFQNLGK_vs_SPRL1_VLTHSELAPLR
38 и 140
0,020
0,767
ENPP2_TYLHTYESEI_vs_PGRP2_AGLLRPDYALLGHR
83 и 126
0,043
0,732
AFAM_HFQNLGK_vs_VTDB_ELPEHTVK
38 и 147
0,010
0,797
ENPP2_TYLHTYESEI_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
83 и 135
0,037
0,740
ANGT_DPTFIPAPIQAK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
42 и 135
0,047
0,729
ENPP2_TYLHTYESEI_vs_SHBG_IALGGLLFPASNLR
83 и 18
0,020
0,767
ANGT_DPTFIPAPIQAK_vs_SHBG_IALGGLLFPASNLR
42 и 18
0,012
0,788
ENPP2_TYLHTYESEI_vs_SOM2.CSH_SVEGSCGF
83 и 139
0,042
0,734
APOH_ATVVYQGER_vs_IBP1_VVESLAK
48 и 97
0,036
0,741
F13B_GDTYPAELYITGSILR_vs_IBP1_VVESLAK
84 и 97
0,048
0,727
APOH_ATVVYQGER_vs_PGRP2_AGLLRPDYALLGHR
48 и 126
0,033
0,745
F13B_GDTYPAELYITGSILR_vs_IBP2_LIQGAPTIR
84 и 98
0,020
0,767
APOH_ATVVYQGER_vs_SHBG_IALGGLLFPASNLR
48 и 18
0,020
0,767
F13B_GDTYPAELYITGSILR_vs_LYAM1_SYYWIGIR
84 и 120
0,036
0,741
B2MG_VEHSDLSFSK_vs_IBP2_LIQGAPTIR
50 и 98
0,042
0,734
F13B_GDTYPAELYITGSILR_vs_PGRP2_AGLLRPDYALLGHR
84 и 126
0,047
0,729
B2MG_VEHSDLSFSK_vs_SHBG_IALGGLLFPASNLR
50 и 18
0,026
0,756
F13B_GDTYPAELYITGSILR_vs_SHBG_IALGGLLFPASNLR
84 и 18
0,011
0,794
B2MG_VEHSDLSFSK_vs_SOM2.CSH_SVEGSCGF
50 и 139
0,040
0,736
F13B_GDTYPAELYITGSILR_vs_TENX_LSQLSVTDVTTSSLR
84 и 142
0,040
0,736
B2MG_VEHSDLSFSK_vs_TENX_LSQLSVTDVTTSSLR
50 и 142
0,030
0,749
FBLN3_IPSNPSHR_vs_TENX_LSQLSVTDVTTSSLR
87 и 142
0,016
0,778
B2MG_VNHVTLSQPK_vs_IBP1_VVESLAK
51 и 97
0,045
0,731
FETUA_FSVVYAK_vs_IBP1_VVESLAK
88 и 97
0,034
0,743
B2MG_VNHVTLSQPK_vs_IBP2_LIQGAPTIR
51 и 98
0,032
0,747
FETUA_FSVVYAK_vs_IBP2_LIQGAPTIR
88 и 98
0,017
0,774
B2MG_VNHVTLSQPK_vs_LYAM1_SYYWIGIR
51 и 120
0,042
0,734
FETUA_FSVVYAK_vs_ITIH4_ILDDLSPR
88 и 112
0,012
0,790
B2MG_VNHVTLSQPK_vs_SHBG_IALGGLLFPASNLR
51 и 18
0,019
0,769
FETUA_FSVVYAK_vs_LYAM1_SYYWIGIR
88 и 120
0,007
0,808
B2MG_VNHVTLSQPK_vs_SOM2.CSH_SVEGSCGF
51 и 139
0,039
0,738
FETUA_FSVVYAK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
88 и 135
0,021
0,765
B2MG_VNHVTLSQPK_vs_TENX_LSQLSVTDVTTSSLR
51 и 142
0,019
0,769
FETUA_FSVVYAK_vs_SHBG_IALGGLLFPASNLR
88 и 18
0,013
0,787
CATD_VGFAEAAR_vs_ALS_IRPHTFTGLSGLR
57 и 40
0,007
0,812
FETUA_FSVVYAK_vs_SOM2.CSH_SVEGSCGF
88 и 139
0,023
0,761
CATD_VGFAEAAR_vs_C163A_INPASLDK
57 и 54
0,001
0,886
FETUA_FSVVYAK_vs_VTDB_ELPEHTVK
88 и 147
0,010
0,797
CATD_VGFAEAAR_vs_CHL1_VIAVNEVGR
57 и 66
0,032
0,747
FETUA_HTLNQIDEVK_vs_IBP1_VVESLAK
89 и 97
0,036
0,741
CATD_VGFAEAAR_vs_CRIS3_AVSPPAR
57 и 78
0,019
0,769
FETUA_HTLNQIDEVK_vs_IBP2_LIQGAPTIR
89 и 98
0,009
0,801
CATD_VGFAEAAR_vs_CRIS3_YEDLYSNCK
57 и 79
0,020
0,767
FETUA_HTLNQIDEVK_vs_ITIH4_ILDDLSPR
89 и 112
0,003
0,837
CATD_VGFAEAAR_vs_CSH_AHQLAIDTYQEFEETYIPK
57 и 80
0,001
0,866
FETUA_HTLNQIDEVK_vs_LYAM1_SYYWIGIR
89 и 120
0,005
0,823
CATD_VGFAEAAR_vs_CSH_ISLLLIESWLEPVR
57 и 81
0,001
0,868
FETUA_HTLNQIDEVK_vs_PGRP2_AGLLRPDYALLGHR
89 и 126
0,016
0,778
CATD_VGFAEAAR_vs_FBLN1_TGYYFDGISR
57 и 86
0,010
0,796
FETUA_HTLNQIDEVK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
89 и 135
0,026
0,756
CATD_VGFAEAAR_vs_IBP1_VVESLAK
57 и 97
0,001
0,870
FETUA_HTLNQIDEVK_vs_SHBG_IALGGLLFPASNLR
89 и 18
0,003
0,844
CATD_VGFAEAAR_vs_IBP2_LIQGAPTIR
57 и 98
0,001
0,868
FETUA_HTLNQIDEVK_vs_SOM2.CSH_SVEGSCGF
89 и 139
0,021
0,765
CATD_VGFAEAAR_vs_IBP3_FLNVLSPR
57 и 99
0,029
0,750
FETUA_HTLNQIDEVK_vs_SPRL1_VLTHSELAPLR
89 и 140
0,019
0,769
CATD_VGFAEAAR_vs_IBP3_YGQPLPGYTTK
57 и 100
0,016
0,776
FETUA_HTLNQIDEVK_vs_VTDB_ELPEHTVK
89 и 147
0,004
0,830
CATD_VGFAEAAR_vs_IGF2_GIVEECCFR
57 и 103
0,030
0,749
HEMO_NFPSPVDAAFR_vs_SHBG_IALGGLLFPASNLR
93 и 18
0,021
0,765
CATD_VGFAEAAR_vs_ITIH4_ILDDLSPR
57 и 112
0,003
0,837
HEMO_NFPSPVDAAFR_vs_TENX_LSQLSVTDVTTSSLR
93 и 142
0,023
0,761
CATD_VGFAEAAR_vs_LYAM1_SYYWIGIR
57 и 120
0,001
0,866
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,023
0,761
CATD_VGFAEAAR_vs_NCAM1_GLGEISAASEFK
57 и 121
0,008
0,807
IBP4_QCHPALDGQR_vs_TENX_LSQLSVTDVTTSSLR
2 и 142
0,018
0,772
CATD_VGFAEAAR_vs_PGRP2_AGLLRPDYALLGHR
57 и 126
0,001
0,892
IBP6_GAQTLYVPNCDHR_vs_TENX_LSQLSVTDVTTSSLR
101 и 142
0,008
0,805
CATD_VGFAEAAR_vs_PRG2_WNFAYWAAHQPWSR
57 и 129
0,008
0,805
IBP6_HLDSVLQQLQTEVYR_vs_SHBG_IALGGLLFPASNLR
102 и 18
0,042
0,734
CATD_VGFAEAAR_vs_PSG1_FQLPGQK
57 и 131
0,013
0,785
IBP6_HLDSVLQQLQTEVYR_vs_TENX_LSQLSVTDVTTSSLR
102 и 142
0,006
0,816
CATD_VGFAEAAR_vs_PSG3_VSAPSGTGHLPGLNPL
57 и 134
0,016
0,778
ITIH3_ALDLSLK_vs_IBP1_VVESLAK
111 и 97
0,032
0,747
CATD_VGFAEAAR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
57 и 135
0,003
0,846
ITIH3_ALDLSLK_vs_IBP2_LIQGAPTIR
111 и 98
0,019
0,770
CATD_VGFAEAAR_vs_PSG9_LFIPQITR
57 и 136
0,009
0,801
ITIH3_ALDLSLK_vs_SHBG_IALGGLLFPASNLR
111 и 18
0,006
0,816
CATD_VGFAEAAR_vs_SHBG_IALGGLLFPASNLR
57 и 18
0,003
0,841
ITIH4_NPLVWVHASPEHVVVTR_vs_ALS_IRPHTFTGLSGLR
113 и 40
0,028
0,753
CATD_VGFAEAAR_vs_SOM2.CSH_NYGLLYCFR
57 и 138
0,001
0,868
ITIH4_NPLVWVHASPEHVVVTR_vs_CSH_AHQLAIDTYQEFEETYIPK
113 и 80
0,043
0,733
CATD_VGFAEAAR_vs_SOM2.CSH_SVEGSCGF
57 и 139
0,001
0,875
ITIH4_NPLVWVHASPEHVVVTR_vs_FBLN1_TGYYFDGISR
113 и 86
0,019
0,769
CATD_VGFAEAAR_vs_SPRL1_VLTHSELAPLR
57 и 140
0,002
0,848
ITIH4_NPLVWVHASPEHVVVTR_vs_IBP3_FLNVLSPR
113 и 99
0,009
0,799
CATD_VGFAEAAR_vs_TENX_LNWEAPPGAFDSFLLR
57 и 141
0,017
0,774
ITIH4_NPLVWVHASPEHVVVTR_vs_IBP3_YGQPLPGYTTK
113 и 100
0,023
0,762
CATD_VGFAEAAR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
57 и 144
0,014
0,783
ITIH4_NPLVWVHASPEHVVVTR_vs_IGF2_GIVEECCFR
113 и 103
0,010
0,795
CATD_VGFAEAAR_vs_VTDB_ELPEHTVK
57 и 147
0,002
0,848
ITIH4_NPLVWVHASPEHVVVTR_vs_ITIH4_ILDDLSPR
113 и 112
0,048
0,727
CATD_VSTLPAITLK_vs_ALS_IRPHTFTGLSGLR
58 и 40
0,001
0,866
ITIH4_NPLVWVHASPEHVVVTR_vs_NCAM1_GLGEISAASEFK
113 и 121
0,033
0,745
CATD_VSTLPAITLK_vs_C163A_INPASLDK
58 и 54
0,003
0,846
ITIH4_NPLVWVHASPEHVVVTR_vs_PSG3_VSAPSGTGHLPGLNPL
113 и 134
0,010
0,797
CATD_VSTLPAITLK_vs_CHL1_VIAVNEVGR
58 и 66
0,010
0,796
ITIH4_NPLVWVHASPEHVVVTR_vs_SOM2.CSH_NYGLLYCFR
113 и 138
0,036
0,742
CATD_VSTLPAITLK_vs_CRIS3_AVSPPAR
58 и 78
0,013
0,787
ITIH4_NPLVWVHASPEHVVVTR_vs_SPRL1_VLTHSELAPLR
113 и 140
0,038
0,738
CATD_VSTLPAITLK_vs_CRIS3_YEDLYSNCK
58 и 79
0,011
0,794
ITIH4_NPLVWVHASPEHVVVTR_vs_TENX_LNWEAPPGAFDSFLLR
113 и 141
0,021
0,766
CATD_VSTLPAITLK_vs_CSH_AHQLAIDTYQEFEETYIPK
58 и 80
0,001
0,886
ITIH4_NPLVWVHASPEHVVVTR_vs_TENX_LSQLSVTDVTTSSLR
113 и 142
0,003
0,842
CATD_VSTLPAITLK_vs_CSH_ISLLLIESWLEPVR
58 и 81
0,001
0,895
ITIH4_NPLVWVHASPEHVVVTR_vs_VTDB_ELPEHTVK
113 и 147
0,021
0,766
CATD_VSTLPAITLK_vs_FBLN1_TGYYFDGISR
58 и 86
0,003
0,843
ITIH4_QLGLPGPPDVPDHAAYHPF_vs_TENX_LSQLSVTDVTTSSLR
114 и 142
0,037
0,740
CATD_VSTLPAITLK_vs_IBP1_VVESLAK
58 и 97
0,000
0,906
KNG1_DIPTNSPELEETLTHTITK_vs_TENX_LSQLSVTDVTTSSLR
116 и 142
0,020
0,767
CATD_VSTLPAITLK_vs_IBP2_LIQGAPTIR
58 и 98
0,001
0,882
KNG1_QVVAGLNFR_vs_SHBG_IALGGLLFPASNLR
117 и 18
0,028
0,752
CATD_VSTLPAITLK_vs_IBP3_FLNVLSPR
58 и 99
0,009
0,799
KNG1_QVVAGLNFR_vs_TENX_LSQLSVTDVTTSSLR
117 и 142
0,021
0,765
CATD_VSTLPAITLK_vs_IBP3_YGQPLPGYTTK
58 и 100
0,010
0,797
LBP_ITGFLKPGK_vs_TENX_LSQLSVTDVTTSSLR
118 и 142
0,018
0,772
CATD_VSTLPAITLK_vs_IGF2_GIVEECCFR
58 и 103
0,016
0,778
PAPP1_DIPHWLNPTR_vs_IBP1_VVESLAK
122 и 97
0,037
0,740
CATD_VSTLPAITLK_vs_ITIH4_ILDDLSPR
58 и 112
0,001
0,870
PAPP1_DIPHWLNPTR_vs_IBP2_LIQGAPTIR
122 и 98
0,023
0,761
CATD_VSTLPAITLK_vs_LYAM1_SYYWIGIR
58 и 120
0,001
0,888
PAPP1_DIPHWLNPTR_vs_PGRP2_AGLLRPDYALLGHR
122 и 126
0,037
0,740
CATD_VSTLPAITLK_vs_NCAM1_GLGEISAASEFK
58 и 121
0,004
0,826
PAPP1_DIPHWLNPTR_vs_SHBG_IALGGLLFPASNLR
122 и 18
0,033
0,745
CATD_VSTLPAITLK_vs_PGRP2_AGLLRPDYALLGHR
58 и 126
0,000
0,926
PAPP1_DIPHWLNPTR_vs_SOM2.CSH_SVEGSCGF
122 и 139
0,010
0,797
CATD_VSTLPAITLK_vs_PRG2_WNFAYWAAHQPWSR
58 и 129
0,016
0,776
PEDF_LQSLFDSPDFSK_vs_IBP1_VVESLAK
124 и 97
0,036
0,741
CATD_VSTLPAITLK_vs_PSG1_FQLPGQK
58 и 131
0,013
0,787
PEDF_LQSLFDSPDFSK_vs_IBP2_LIQGAPTIR
124 и 98
0,040
0,736
CATD_VSTLPAITLK_vs_PSG3_VSAPSGTGHLPGLNPL
58 и 134
0,006
0,814
PEDF_LQSLFDSPDFSK_vs_ITIH4_ILDDLSPR
124 и 112
0,025
0,758
CATD_VSTLPAITLK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
58 и 135
0,004
0,830
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,028
0,752
CATD_VSTLPAITLK_vs_PSG9_LFIPQITR
58 и 136
0,010
0,797
PEDF_LQSLFDSPDFSK_vs_PGRP2_AGLLRPDYALLGHR
124 и 126
0,048
0,727
CATD_VSTLPAITLK_vs_SHBG_IALGGLLFPASNLR
58 и 18
0,001
0,875
PEDF_LQSLFDSPDFSK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
124 и 135
0,045
0,731
CATD_VSTLPAITLK_vs_SOM2.CSH_NYGLLYCFR
58 и 138
0,001
0,875
PEDF_LQSLFDSPDFSK_vs_SHBG_IALGGLLFPASNLR
124 и 18
0,011
0,794
CATD_VSTLPAITLK_vs_SOM2.CSH_SVEGSCGF
58 и 139
0,001
0,886
PEDF_LQSLFDSPDFSK_vs_VTDB_ELPEHTVK
124 и 147
0,030
0,749
CATD_VSTLPAITLK_vs_SPRL1_VLTHSELAPLR
58 и 140
0,001
0,868
PEDF_TVQAVLTVPK_vs_SHBG_IALGGLLFPASNLR
125 и 18
0,024
0,759
CATD_VSTLPAITLK_vs_TENX_LNWEAPPGAFDSFLLR
58 и 141
0,005
0,821
PEDF_TVQAVLTVPK_vs_TENX_LSQLSVTDVTTSSLR
125 и 142
0,020
0,767
CATD_VSTLPAITLK_vs_TENX_LSQLSVTDVTTSSLR
58 и 142
0,028
0,752
PSG11_LFIPQITPK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
132 и 135
0,026
0,756
CATD_VSTLPAITLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
58 и 144
0,006
0,816
PSG11_LFIPQITPK_vs_SOM2.CSH_SVEGSCGF
132 и 139
0,028
0,752
CATD_VSTLPAITLK_vs_VTDB_ELPEHTVK
58 и 147
0,002
0,863
PTGDS_GPGEDFR_vs_SHBG_IALGGLLFPASNLR
137 и 18
0,030
0,749
CBPN_EALIQFLEQVHQGIK_vs_SHBG_IALGGLLFPASNLR
59 и 18
0,014
0,783
PTGDS_GPGEDFR_vs_SOM2.CSH_SVEGSCGF
137 и 139
0,042
0,734
CBPN_EALIQFLEQVHQGIK_vs_TENX_LSQLSVTDVTTSSLR
59 и 142
0,043
0,732
THBG_AVLHIGEK_vs_SHBG_IALGGLLFPASNLR
143 и 18
0,040
0,736
CBPN_NNANGVDLNR_vs_SHBG_IALGGLLFPASNLR
60 и 18
0,011
0,794
THBG_AVLHIGEK_vs_TENX_LSQLSVTDVTTSSLR
143 и 142
0,025
0,758
CD14_LTVGAAQVPAQLLVGALR_vs_PGRP2_AGLLRPDYALLGHR
61 и 126
0,048
0,727
VTNC_GQYCYELDEK_vs_ITIH4_ILDDLSPR
149 и 112
0,026
0,756
CD14_LTVGAAQVPAQLLVGALR_vs_SHBG_IALGGLLFPASNLR
61 и 18
0,017
0,774
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,029
0,750
CD14_LTVGAAQVPAQLLVGALR_vs_TENX_LSQLSVTDVTTSSLR
61 и 142
0,037
0,740
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,010
0,797
CD14_SWLAELQQWLKPGLK_vs_IBP1_VVESLAK
62 и 97
0,047
0,729
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,018
0,772
Таблица 43: эффективность классификации по обращениям, недели с 17 до 21.
AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, без BMI-стратификации.
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
A2GL_DLLLPQPDLR_vs_PSG3_VSAPSGTGHLPGLNPL
34 и 134
0,002
0,609
HABP2_FLNWIK_vs_CHL1_VIAVNEVGR
92 и 66
0,003
0,607
A2GL_DLLLPQPDLR_vs_SHBG_IALGGLLFPASNLR
34 и 18
0,002
0,610
HABP2_FLNWIK_vs_IGF2_GIVEECCFR
92 и 103
0,000
0,628
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,001
0,614
HABP2_FLNWIK_vs_PSG3_VSAPSGTGHLPGLNPL
92 и 134
0,001
0,623
AFAM_HFQNLGK_vs_IBP3_YGQPLPGYTTK
38 и 100
0,003
0,605
HABP2_FLNWIK_vs_SHBG_IALGGLLFPASNLR
92 и 18
0,003
0,607
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,001
0,620
HABP2_FLNWIK_vs_TENX_LNWEAPPGAFDSFLLR
92 и 141
0,001
0,617
AFAM_HFQNLGK_vs_PSG3_VSAPSGTGHLPGLNPL
38 и 134
0,003
0,608
HABP2_FLNWIK_vs_TENX_LSQLSVTDVTTSSLR
92 и 142
0,004
0,601
APOC3_GWVTDGFSSLK_vs_ALS_IRPHTFTGLSGLR
47 и 40
0,001
0,622
HEMO_NFPSPVDAAFR_vs_PSG3_VSAPSGTGHLPGLNPL
93 и 134
0,005
0,600
APOC3_GWVTDGFSSLK_vs_C163A_INPASLDK
47 и 54
0,001
0,623
HEMO_NFPSPVDAAFR_vs_SHBG_IALGGLLFPASNLR
93 и 18
0,001
0,614
APOC3_GWVTDGFSSLK_vs_CHL1_VIAVNEVGR
47 и 66
0,001
0,618
HEMO_NFPSPVDAAFR_vs_TENX_LNWEAPPGAFDSFLLR
93 и 141
0,004
0,601
APOC3_GWVTDGFSSLK_vs_CRIS3_AVSPPAR
47 и 78
0,001
0,618
IBP4_QCHPALDGQR_vs_CHL1_VIAVNEVGR
2 и 66
0,002
0,610
APOC3_GWVTDGFSSLK_vs_CRIS3_YEDLYSNCK
47 и 79
0,001
0,623
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,004
0,601
APOC3_GWVTDGFSSLK_vs_CSH_AHQLAIDTYQEFEETYIPK
47 и 80
0,001
0,624
IBP4_QCHPALDGQR_vs_IGF2_GIVEECCFR
2 и 103
0,000
0,626
APOC3_GWVTDGFSSLK_vs_CSH_ISLLLIESWLEPVR
47 и 81
0,002
0,610
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,003
0,606
APOC3_GWVTDGFSSLK_vs_FBLN1_TGYYFDGISR
47 и 86
0,001
0,618
IBP4_QCHPALDGQR_vs_PGRP2_AGLLRPDYALLGHR
2 и 126
0,000
0,625
APOC3_GWVTDGFSSLK_vs_IBP2_LIQGAPTIR
47 и 98
0,004
0,602
IBP4_QCHPALDGQR_vs_PSG3_VSAPSGTGHLPGLNPL
2 и 134
0,000
0,644
APOC3_GWVTDGFSSLK_vs_IBP3_FLNVLSPR
47 и 99
0,000
0,633
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,000
0,635
APOC3_GWVTDGFSSLK_vs_IBP3_YGQPLPGYTTK
47 и 100
0,000
0,632
IBP4_QCHPALDGQR_vs_TENX_LNWEAPPGAFDSFLLR
2 и 141
0,001
0,619
APOC3_GWVTDGFSSLK_vs_IGF2_GIVEECCFR
47 и 103
0,000
0,640
INHBC_LDFHFSSDR_vs_CHL1_VIAVNEVGR
107 и 66
0,002
0,613
APOC3_GWVTDGFSSLK_vs_ITIH4_ILDDLSPR
47 и 112
0,001
0,623
INHBC_LDFHFSSDR_vs_IBP3_FLNVLSPR
107 и 99
0,001
0,614
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,000
0,633
INHBC_LDFHFSSDR_vs_IBP3_YGQPLPGYTTK
107 и 100
0,001
0,617
APOC3_GWVTDGFSSLK_vs_NCAM1_GLGEISAASEFK
47 и 121
0,001
0,618
INHBC_LDFHFSSDR_vs_IGF2_GIVEECCFR
107 и 103
0,000
0,637
APOC3_GWVTDGFSSLK_vs_PGRP2_AGLLRPDYALLGHR
47 и 126
0,000
0,630
INHBC_LDFHFSSDR_vs_ITIH4_ILDDLSPR
107 и 112
0,005
0,600
APOC3_GWVTDGFSSLK_vs_PRG2_WNFAYWAAHQPWSR
47 и 129
0,001
0,622
INHBC_LDFHFSSDR_vs_LYAM1_SYYWIGIR
107 и 120
0,004
0,601
APOC3_GWVTDGFSSLK_vs_PSG1_FQLPGQK
47 и 131
0,004
0,603
INHBC_LDFHFSSDR_vs_PGRP2_AGLLRPDYALLGHR
107 и 126
0,001
0,621
APOC3_GWVTDGFSSLK_vs_PSG3_VSAPSGTGHLPGLNPL
47 и 134
0,000
0,644
INHBC_LDFHFSSDR_vs_PRG2_WNFAYWAAHQPWSR
107 и 129
0,003
0,605
APOC3_GWVTDGFSSLK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
47 и 135
0,003
0,607
INHBC_LDFHFSSDR_vs_PSG3_VSAPSGTGHLPGLNPL
107 и 134
0,000
0,636
APOC3_GWVTDGFSSLK_vs_PSG9_LFIPQITR
47 и 136
0,004
0,601
INHBC_LDFHFSSDR_vs_SHBG_IALGGLLFPASNLR
107 и 18
0,001
0,617
APOC3_GWVTDGFSSLK_vs_SHBG_IALGGLLFPASNLR
47 и 18
0,000
0,630
INHBC_LDFHFSSDR_vs_TENX_LNWEAPPGAFDSFLLR
107 и 141
0,000
0,627
APOC3_GWVTDGFSSLK_vs_SOM2.CSH_NYGLLYCFR
47 и 138
0,003
0,605
INHBC_LDFHFSSDR_vs_TENX_LSQLSVTDVTTSSLR
107 и 142
0,002
0,610
APOC3_GWVTDGFSSLK_vs_SOM2.CSH_SVEGSCGF
47 и 139
0,001
0,616
INHBC_LDFHFSSDR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
107 и 144
0,003
0,606
APOC3_GWVTDGFSSLK_vs_SPRL1_VLTHSELAPLR
47 и 140
0,002
0,609
INHBC_LDFHFSSDR_vs_VTDB_ELPEHTVK
107 и 147
0,001
0,613
APOC3_GWVTDGFSSLK_vs_TENX_LNWEAPPGAFDSFLLR
47 и 141
0,000
0,631
ITIH3_ALDLSLK_vs_SHBG_IALGGLLFPASNLR
111 и 18
0,000
0,625
APOC3_GWVTDGFSSLK_vs_TENX_LSQLSVTDVTTSSLR
47 и 142
0,001
0,624
KNG1_DIPTNSPELEETLTHTITK_vs_PSG3_VSAPSGTGHLPGLNPL
116 и 134
0,002
0,609
APOC3_GWVTDGFSSLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
47 и 144
0,000
0,628
KNG1_DIPTNSPELEETLTHTITK_vs_SHBG_IALGGLLFPASNLR
116 и 18
0,004
0,601
APOC3_GWVTDGFSSLK_vs_VTDB_ELPEHTVK
47 и 147
0,000
0,627
KNG1_QVVAGLNFR_vs_SHBG_IALGGLLFPASNLR
117 и 18
0,005
0,601
APOH_ATVVYQGER_vs_IGF2_GIVEECCFR
48 и 103
0,002
0,613
LBP_ITGFLKPGK_vs_CHL1_VIAVNEVGR
118 и 66
0,004
0,602
APOH_ATVVYQGER_vs_PSG3_VSAPSGTGHLPGLNPL
48 и 134
0,002
0,612
LBP_ITGFLKPGK_vs_CRIS3_YEDLYSNCK
118 и 79
0,003
0,606
APOH_ATVVYQGER_vs_SHBG_IALGGLLFPASNLR
48 и 18
0,003
0,606
LBP_ITGFLKPGK_vs_IGF2_GIVEECCFR
118 и 103
0,002
0,612
B2MG_VNHVTLSQPK_vs_IGF2_GIVEECCFR
51 и 103
0,003
0,607
LBP_ITGFLKPGK_vs_LYAM1_SYYWIGIR
118 и 120
0,003
0,607
B2MG_VNHVTLSQPK_vs_PSG3_VSAPSGTGHLPGLNPL
51 и 134
0,004
0,603
LBP_ITGFLKPGK_vs_PGRP2_AGLLRPDYALLGHR
118 и 126
0,003
0,605
B2MG_VNHVTLSQPK_vs_SHBG_IALGGLLFPASNLR
51 и 18
0,002
0,609
LBP_ITGFLKPGK_vs_PSG3_VSAPSGTGHLPGLNPL
118 и 134
0,000
0,638
C1QB_VPGLYYFTYHASSR_vs_IGF2_GIVEECCFR
55 и 103
0,002
0,613
LBP_ITGFLKPGK_vs_SHBG_IALGGLLFPASNLR
118 и 18
0,001
0,621
C1QB_VPGLYYFTYHASSR_vs_PGRP2_AGLLRPDYALLGHR
55 и 126
0,005
0,601
LBP_ITGFLKPGK_vs_TENX_LNWEAPPGAFDSFLLR
118 и 141
0,005
0,600
C1QB_VPGLYYFTYHASSR_vs_PRG2_WNFAYWAAHQPWSR
55 и 129
0,005
0,601
LBP_ITGFLKPGK_vs_VTDB_ELPEHTVK
118 и 147
0,004
0,601
C1QB_VPGLYYFTYHASSR_vs_PSG3_VSAPSGTGHLPGLNPL
55 и 134
0,003
0,605
LBP_ITLPDFTGDLR_vs_C163A_INPASLDK
119 и 54
0,002
0,608
C1QB_VPGLYYFTYHASSR_vs_SHBG_IALGGLLFPASNLR
55 и 18
0,001
0,616
LBP_ITLPDFTGDLR_vs_CHL1_VIAVNEVGR
119 и 66
0,001
0,619
C1QB_VPGLYYFTYHASSR_vs_TENX_LNWEAPPGAFDSFLLR
55 и 141
0,003
0,605
LBP_ITLPDFTGDLR_vs_CRIS3_AVSPPAR
119 и 78
0,001
0,615
CATD_VGFAEAAR_vs_PSG3_VSAPSGTGHLPGLNPL
57 и 134
0,002
0,612
LBP_ITLPDFTGDLR_vs_CRIS3_YEDLYSNCK
119 и 79
0,001
0,620
CATD_VGFAEAAR_vs_SHBG_IALGGLLFPASNLR
57 и 18
0,005
0,600
LBP_ITLPDFTGDLR_vs_CSH_AHQLAIDTYQEFEETYIPK
119 и 80
0,004
0,603
CATD_VGFAEAAR_vs_TENX_LNWEAPPGAFDSFLLR
57 и 141
0,003
0,606
LBP_ITLPDFTGDLR_vs_IBP3_FLNVLSPR
119 и 99
0,003
0,604
CATD_VSTLPAITLK_vs_PSG3_VSAPSGTGHLPGLNPL
58 и 134
0,003
0,606
LBP_ITLPDFTGDLR_vs_IBP3_YGQPLPGYTTK
119 и 100
0,002
0,610
CATD_VSTLPAITLK_vs_SHBG_IALGGLLFPASNLR
58 и 18
0,005
0,600
LBP_ITLPDFTGDLR_vs_IGF2_GIVEECCFR
119 и 103
0,000
0,629
CATD_VSTLPAITLK_vs_TENX_LNWEAPPGAFDSFLLR
58 и 141
0,005
0,601
LBP_ITLPDFTGDLR_vs_ITIH4_ILDDLSPR
119 и 112
0,002
0,608
CD14_LTVGAAQVPAQLLVGALR_vs_IGF2_GIVEECCFR
61 и 103
0,004
0,604
LBP_ITLPDFTGDLR_vs_LYAM1_SYYWIGIR
119 и 120
0,000
0,625
CD14_LTVGAAQVPAQLLVGALR_vs_PSG3_VSAPSGTGHLPGLNPL
61 и 134
0,003
0,605
LBP_ITLPDFTGDLR_vs_NCAM1_GLGEISAASEFK
119 и 121
0,003
0,606
CD14_LTVGAAQVPAQLLVGALR_vs_SHBG_IALGGLLFPASNLR
61 и 18
0,003
0,605
LBP_ITLPDFTGDLR_vs_PGRP2_AGLLRPDYALLGHR
119 и 126
0,001
0,622
CD14_LTVGAAQVPAQLLVGALR_vs_TENX_LNWEAPPGAFDSFLLR
61 и 141
0,004
0,602
LBP_ITLPDFTGDLR_vs_PRG2_WNFAYWAAHQPWSR
119 и 129
0,002
0,611
CD14_SWLAELQQWLKPGLK_vs_PSG3_VSAPSGTGHLPGLNPL
62 и 134
0,004
0,604
LBP_ITLPDFTGDLR_vs_PSG3_VSAPSGTGHLPGLNPL
119 и 134
0,000
0,651
CD14_SWLAELQQWLKPGLK_vs_SHBG_IALGGLLFPASNLR
62 и 18
0,002
0,609
LBP_ITLPDFTGDLR_vs_SHBG_IALGGLLFPASNLR
119 и 18
0,000
0,636
CD14_SWLAELQQWLKPGLK_vs_TENX_LNWEAPPGAFDSFLLR
62 и 141
0,004
0,602
LBP_ITLPDFTGDLR_vs_SOM2.CSH_SVEGSCGF
119 и 139
0,003
0,608
CFAB_YGLVTYATYPK_vs_CHL1_VIAVNEVGR
64 и 66
0,002
0,608
LBP_ITLPDFTGDLR_vs_TENX_LNWEAPPGAFDSFLLR
119 и 141
0,001
0,619
CFAB_YGLVTYATYPK_vs_IGF2_GIVEECCFR
64 и 103
0,001
0,623
LBP_ITLPDFTGDLR_vs_TENX_LSQLSVTDVTTSSLR
119 и 142
0,003
0,606
CFAB_YGLVTYATYPK_vs_LYAM1_SYYWIGIR
64 и 120
0,004
0,603
LBP_ITLPDFTGDLR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
119 и 144
0,002
0,610
CFAB_YGLVTYATYPK_vs_PGRP2_AGLLRPDYALLGHR
64 и 126
0,003
0,608
LBP_ITLPDFTGDLR_vs_VTDB_ELPEHTVK
119 и 147
0,000
0,626
CFAB_YGLVTYATYPK_vs_PSG3_VSAPSGTGHLPGLNPL
64 и 134
0,000
0,632
PEDF_LQSLFDSPDFSK_vs_IGF2_GIVEECCFR
124 и 103
0,004
0,602
CFAB_YGLVTYATYPK_vs_SHBG_IALGGLLFPASNLR
64 и 18
0,001
0,621
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,005
0,601
CFAB_YGLVTYATYPK_vs_TENX_LNWEAPPGAFDSFLLR
64 и 141
0,002
0,611
PEDF_LQSLFDSPDFSK_vs_PSG3_VSAPSGTGHLPGLNPL
124 и 134
0,001
0,617
CFAB_YGLVTYATYPK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
64 и 144
0,001
0,613
PEDF_LQSLFDSPDFSK_vs_SHBG_IALGGLLFPASNLR
124 и 18
0,004
0,603
CFAB_YGLVTYATYPK_vs_VTDB_ELPEHTVK
64 и 147
0,004
0,601
PEDF_LQSLFDSPDFSK_vs_TENX_LNWEAPPGAFDSFLLR
124 и 141
0,001
0,614
CO5_TLLPVSKPEIR_vs_IGF2_GIVEECCFR
70 и 103
0,004
0,603
PEDF_LQSLFDSPDFSK_vs_TENX_LSQLSVTDVTTSSLR
124 и 142
0,004
0,602
CO5_TLLPVSKPEIR_vs_PSG3_VSAPSGTGHLPGLNPL
70 и 134
0,001
0,618
PEDF_TVQAVLTVPK_vs_IGF2_GIVEECCFR
125 и 103
0,003
0,608
CO5_TLLPVSKPEIR_vs_SHBG_IALGGLLFPASNLR
70 и 18
0,002
0,610
PEDF_TVQAVLTVPK_vs_PSG3_VSAPSGTGHLPGLNPL
125 и 134
0,002
0,613
CO5_TLLPVSKPEIR_vs_TENX_LNWEAPPGAFDSFLLR
70 и 141
0,004
0,602
PEDF_TVQAVLTVPK_vs_TENX_LNWEAPPGAFDSFLLR
125 и 141
0,002
0,610
CO5_VFQFLEK_vs_PSG3_VSAPSGTGHLPGLNPL
71 и 134
0,001
0,614
VTNC_GQYCYELDEK_vs_ALS_IRPHTFTGLSGLR
149 и 40
0,005
0,600
CO5_VFQFLEK_vs_SHBG_IALGGLLFPASNLR
71 и 18
0,002
0,612
VTNC_GQYCYELDEK_vs_CHL1_VIAVNEVGR
149 и 66
0,002
0,613
CO5_VFQFLEK_vs_TENX_LNWEAPPGAFDSFLLR
71 и 141
0,004
0,602
VTNC_GQYCYELDEK_vs_IBP3_FLNVLSPR
149 и 99
0,002
0,609
CO6_ALNHLPLEYNSALYSR_vs_SHBG_IALGGLLFPASNLR
72 и 18
0,004
0,603
VTNC_GQYCYELDEK_vs_IBP3_YGQPLPGYTTK
149 и 100
0,001
0,618
CO8A_SLLQPNK_vs_IGF2_GIVEECCFR
74 и 103
0,001
0,617
VTNC_GQYCYELDEK_vs_IGF2_GIVEECCFR
149 и 103
0,000
0,636
CO8A_SLLQPNK_vs_PSG3_VSAPSGTGHLPGLNPL
74 и 134
0,002
0,613
VTNC_GQYCYELDEK_vs_ITIH4_ILDDLSPR
149 и 112
0,004
0,603
CO8A_SLLQPNK_vs_SHBG_IALGGLLFPASNLR
74 и 18
0,002
0,608
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,001
0,617
CO8A_SLLQPNK_vs_TENX_LNWEAPPGAFDSFLLR
74 и 141
0,002
0,612
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,001
0,621
CO8B_QALEEFQK_vs_IGF2_GIVEECCFR
76 и 103
0,001
0,620
VTNC_GQYCYELDEK_vs_PRG2_WNFAYWAAHQPWSR
149 и 129
0,003
0,608
CO8B_QALEEFQK_vs_PSG3_VSAPSGTGHLPGLNPL
76 и 134
0,001
0,619
VTNC_GQYCYELDEK_vs_PSG3_VSAPSGTGHLPGLNPL
149 и 134
0,000
0,646
CO8B_QALEEFQK_vs_SHBG_IALGGLLFPASNLR
76 и 18
0,003
0,606
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,000
0,640
CO8B_QALEEFQK_vs_TENX_LNWEAPPGAFDSFLLR
76 и 141
0,001
0,616
VTNC_GQYCYELDEK_vs_TENX_LNWEAPPGAFDSFLLR
149 и 141
0,001
0,619
CO8B_QALEEFQK_vs_TENX_LSQLSVTDVTTSSLR
76 и 142
0,003
0,606
VTNC_GQYCYELDEK_vs_TENX_LSQLSVTDVTTSSLR
149 и 142
0,004
0,604
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PSG3_VSAPSGTGHLPGLNPL
82 и 134
0,003
0,604
VTNC_GQYCYELDEK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
149 и 144
0,002
0,609
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_SHBG_IALGGLLFPASNLR
82 и 18
0,001
0,617
VTNC_GQYCYELDEK_vs_VTDB_ELPEHTVK
149 и 147
0,000
0,630
ENPP2_TYLHTYESEI_vs_IGF2_GIVEECCFR
83 и 103
0,004
0,602
VTNC_VDTVDPPYPR_vs_CHL1_VIAVNEVGR
150 и 66
0,001
0,615
ENPP2_TYLHTYESEI_vs_LYAM1_SYYWIGIR
83 и 120
0,004
0,602
VTNC_VDTVDPPYPR_vs_IBP3_YGQPLPGYTTK
150 и 100
0,003
0,606
ENPP2_TYLHTYESEI_vs_PSG3_VSAPSGTGHLPGLNPL
83 и 134
0,004
0,602
VTNC_VDTVDPPYPR_vs_IGF2_GIVEECCFR
150 и 103
0,000
0,631
ENPP2_TYLHTYESEI_vs_SHBG_IALGGLLFPASNLR
83 и 18
0,002
0,611
VTNC_VDTVDPPYPR_vs_ITIH4_ILDDLSPR
150 и 112
0,004
0,604
FBLN3_IPSNPSHR_vs_SHBG_IALGGLLFPASNLR
87 и 18
0,003
0,605
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,001
0,622
FETUA_FSVVYAK_vs_IGF2_GIVEECCFR
88 и 103
0,000
0,629
VTNC_VDTVDPPYPR_vs_NCAM1_GLGEISAASEFK
150 и 121
0,005
0,601
FETUA_FSVVYAK_vs_PSG3_VSAPSGTGHLPGLNPL
88 и 134
0,001
0,617
VTNC_VDTVDPPYPR_vs_PGRP2_AGLLRPDYALLGHR
150 и 126
0,001
0,617
FETUA_FSVVYAK_vs_SHBG_IALGGLLFPASNLR
88 и 18
0,004
0,604
VTNC_VDTVDPPYPR_vs_PRG2_WNFAYWAAHQPWSR
150 и 129
0,004
0,603
FETUA_FSVVYAK_vs_TENX_LNWEAPPGAFDSFLLR
88 и 141
0,003
0,607
VTNC_VDTVDPPYPR_vs_PSG3_VSAPSGTGHLPGLNPL
150 и 134
0,000
0,642
FETUA_HTLNQIDEVK_vs_IGF2_GIVEECCFR
89 и 103
0,002
0,610
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,000
0,632
FETUA_HTLNQIDEVK_vs_PSG3_VSAPSGTGHLPGLNPL
89 и 134
0,002
0,612
VTNC_VDTVDPPYPR_vs_TENX_LNWEAPPGAFDSFLLR
150 и 141
0,001
0,616
FETUA_HTLNQIDEVK_vs_SHBG_IALGGLLFPASNLR
89 и 18
0,004
0,603
VTNC_VDTVDPPYPR_vs_VTDB_ELPEHTVK
150 и 147
0,000
0,642
FETUA_HTLNQIDEVK_vs_TENX_LNWEAPPGAFDSFLLR
89 и 141
0,002
0,612
Таблица 44: эффективность классификации по обращениям, недели с 17 до 21.
AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, с BMI-стратификацией (>22≤37).
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
A2GL_DLLLPQPDLR_vs_CRIS3_AVSPPAR
34 и 78
0,016
0,604
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_SHBG_IALGGLLFPASNLR
82 и 18
0,005
0,622
A2GL_DLLLPQPDLR_vs_CRIS3_YEDLYSNCK
34 и 79
0,008
0,613
ENPP2_TYLHTYESEI_vs_LYAM1_SYYWIGIR
83 и 120
0,013
0,607
A2GL_DLLLPQPDLR_vs_IGF2_GIVEECCFR
34 и 103
0,006
0,617
ENPP2_TYLHTYESEI_vs_SHBG_IALGGLLFPASNLR
83 и 18
0,015
0,605
A2GL_DLLLPQPDLR_vs_LYAM1_SYYWIGIR
34 и 120
0,003
0,629
F13B_GDTYPAELYITGSILR_vs_IGF2_GIVEECCFR
84 и 103
0,009
0,613
A2GL_DLLLPQPDLR_vs_PGRP2_AGLLRPDYALLGHR
34 и 126
0,011
0,609
FETUA_FSVVYAK_vs_IGF2_GIVEECCFR
88 и 103
0,004
0,624
A2GL_DLLLPQPDLR_vs_SHBG_IALGGLLFPASNLR
34 и 18
0,009
0,612
FETUA_HTLNQIDEVK_vs_IGF2_GIVEECCFR
89 и 103
0,012
0,608
AFAM_DADPDTFFAK_vs_IBP3_FLNVLSPR
37 и 99
0,014
0,605
HABP2_FLNWIK_vs_CHL1_VIAVNEVGR
92 и 66
0,014
0,605
AFAM_DADPDTFFAK_vs_IBP3_YGQPLPGYTTK
37 и 100
0,004
0,623
HABP2_FLNWIK_vs_IBP3_FLNVLSPR
92 и 99
0,010
0,610
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,002
0,636
HABP2_FLNWIK_vs_IBP3_YGQPLPGYTTK
92 и 100
0,006
0,618
AFAM_DADPDTFFAK_vs_PSG3_VSAPSGTGHLPGLNPL
37 и 134
0,020
0,600
HABP2_FLNWIK_vs_IGF2_GIVEECCFR
92 и 103
0,001
0,636
AFAM_HFQNLGK_vs_CHL1_VIAVNEVGR
38 и 66
0,018
0,602
HABP2_FLNWIK_vs_LYAM1_SYYWIGIR
92 и 120
0,012
0,608
AFAM_HFQNLGK_vs_IBP3_FLNVLSPR
38 и 99
0,005
0,619
HABP2_FLNWIK_vs_PSG3_VSAPSGTGHLPGLNPL
92 и 134
0,018
0,602
AFAM_HFQNLGK_vs_IBP3_YGQPLPGYTTK
38 и 100
0,002
0,635
HABP2_FLNWIK_vs_TENX_LNWEAPPGAFDSFLLR
92 и 141
0,012
0,608
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,001
0,637
IBP4_QCHPALDGQR_vs_CHL1_VIAVNEVGR
2 и 66
0,013
0,607
AFAM_HFQNLGK_vs_PSG3_VSAPSGTGHLPGLNPL
38 и 134
0,011
0,609
IBP4_QCHPALDGQR_vs_CRIS3_AVSPPAR
2 и 78
0,005
0,621
ANGT_DPTFIPAPIQAK_vs_CHL1_VIAVNEVGR
42 и 66
0,015
0,605
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,002
0,635
ANGT_DPTFIPAPIQAK_vs_CRIS3_AVSPPAR
42 и 78
0,007
0,615
IBP4_QCHPALDGQR_vs_IBP3_FLNVLSPR
2 и 99
0,009
0,611
ANGT_DPTFIPAPIQAK_vs_CRIS3_YEDLYSNCK
42 и 79
0,006
0,618
IBP4_QCHPALDGQR_vs_IBP3_YGQPLPGYTTK
2 и 100
0,003
0,626
ANGT_DPTFIPAPIQAK_vs_IBP3_YGQPLPGYTTK
42 и 100
0,019
0,601
IBP4_QCHPALDGQR_vs_IGF2_GIVEECCFR
2 и 103
0,001
0,646
ANGT_DPTFIPAPIQAK_vs_IGF2_GIVEECCFR
42 и 103
0,008
0,614
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,001
0,640
ANGT_DPTFIPAPIQAK_vs_LYAM1_SYYWIGIR
42 и 120
0,009
0,612
IBP4_QCHPALDGQR_vs_PGRP2_AGLLRPDYALLGHR
2 и 126
0,001
0,637
ANGT_DPTFIPAPIQAK_vs_PGRP2_AGLLRPDYALLGHR
42 и 126
0,016
0,604
IBP4_QCHPALDGQR_vs_PSG3_VSAPSGTGHLPGLNPL
2 и 134
0,005
0,622
ANGT_DPTFIPAPIQAK_vs_PSG3_VSAPSGTGHLPGLNPL
42 и 134
0,012
0,608
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,002
0,634
ANGT_DPTFIPAPIQAK_vs_SHBG_IALGGLLFPASNLR
42 и 18
0,003
0,626
IBP4_QCHPALDGQR_vs_TENX_LNWEAPPGAFDSFLLR
2 и 141
0,008
0,614
ANGT_DPTFIPAPIQAK_vs_SOM2.CSH_SVEGSCGF
42 и 139
0,013
0,607
IBP4_QCHPALDGQR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
2 и 144
0,007
0,615
ANGT_DPTFIPAPIQAK_vs_TENX_LNWEAPPGAFDSFLLR
42 и 141
0,012
0,607
IBP4_QCHPALDGQR_vs_VTDB_ELPEHTVK
2 и 147
0,017
0,603
APOC3_GWVTDGFSSLK_vs_ALS_IRPHTFTGLSGLR
47 и 40
0,010
0,610
INHBC_LDFHFSSDR_vs_ALS_IRPHTFTGLSGLR
107 и 40
0,008
0,614
APOC3_GWVTDGFSSLK_vs_C163A_INPASLDK
47 и 54
0,003
0,628
INHBC_LDFHFSSDR_vs_CHL1_VIAVNEVGR
107 и 66
0,007
0,616
APOC3_GWVTDGFSSLK_vs_CRIS3_AVSPPAR
47 и 78
0,003
0,627
INHBC_LDFHFSSDR_vs_CRIS3_AVSPPAR
107 и 78
0,015
0,604
APOC3_GWVTDGFSSLK_vs_CRIS3_YEDLYSNCK
47 и 79
0,002
0,632
INHBC_LDFHFSSDR_vs_CRIS3_YEDLYSNCK
107 и 79
0,008
0,614
APOC3_GWVTDGFSSLK_vs_IBP3_FLNVLSPR
47 и 99
0,004
0,625
INHBC_LDFHFSSDR_vs_IBP3_FLNVLSPR
107 и 99
0,002
0,634
APOC3_GWVTDGFSSLK_vs_IBP3_YGQPLPGYTTK
47 и 100
0,005
0,621
INHBC_LDFHFSSDR_vs_IBP3_YGQPLPGYTTK
107 и 100
0,001
0,639
APOC3_GWVTDGFSSLK_vs_IGF2_GIVEECCFR
47 и 103
0,003
0,628
INHBC_LDFHFSSDR_vs_IGF2_GIVEECCFR
107 и 103
0,001
0,649
APOC3_GWVTDGFSSLK_vs_ITIH4_ILDDLSPR
47 и 112
0,016
0,603
INHBC_LDFHFSSDR_vs_ITIH4_ILDDLSPR
107 и 112
0,016
0,603
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,002
0,630
INHBC_LDFHFSSDR_vs_LYAM1_SYYWIGIR
107 и 120
0,008
0,613
APOC3_GWVTDGFSSLK_vs_PGRP2_AGLLRPDYALLGHR
47 и 126
0,006
0,617
INHBC_LDFHFSSDR_vs_PGRP2_AGLLRPDYALLGHR
107 и 126
0,005
0,620
APOC3_GWVTDGFSSLK_vs_PSG3_VSAPSGTGHLPGLNPL
47 и 134
0,005
0,621
INHBC_LDFHFSSDR_vs_PSG3_VSAPSGTGHLPGLNPL
107 и 134
0,002
0,635
APOC3_GWVTDGFSSLK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
47 и 135
0,008
0,613
INHBC_LDFHFSSDR_vs_SHBG_IALGGLLFPASNLR
107 и 18
0,006
0,619
APOC3_GWVTDGFSSLK_vs_PSG9_LFIPQITR
47 и 136
0,019
0,601
INHBC_LDFHFSSDR_vs_SPRL1_VLTHSELAPLR
107 и 140
0,006
0,617
APOC3_GWVTDGFSSLK_vs_SHBG_IALGGLLFPASNLR
47 и 18
0,008
0,613
INHBC_LDFHFSSDR_vs_TENX_LNWEAPPGAFDSFLLR
107 и 141
0,001
0,637
APOC3_GWVTDGFSSLK_vs_SOM2.CSH_SVEGSCGF
47 и 139
0,016
0,604
INHBC_LDFHFSSDR_vs_TENX_LSQLSVTDVTTSSLR
107 и 142
0,008
0,614
APOC3_GWVTDGFSSLK_vs_TENX_LNWEAPPGAFDSFLLR
47 и 141
0,009
0,613
INHBC_LDFHFSSDR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
107 и 144
0,010
0,610
APOC3_GWVTDGFSSLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
47 и 144
0,009
0,611
INHBC_LDFHFSSDR_vs_VTDB_ELPEHTVK
107 и 147
0,003
0,627
APOC3_GWVTDGFSSLK_vs_VTDB_ELPEHTVK
47 и 147
0,011
0,609
ITIH3_ALDLSLK_vs_CRIS3_AVSPPAR
111 и 78
0,013
0,607
APOH_ATVVYQGER_vs_IBP3_YGQPLPGYTTK
48 и 100
0,009
0,613
ITIH3_ALDLSLK_vs_CRIS3_YEDLYSNCK
111 и 79
0,009
0,611
APOH_ATVVYQGER_vs_IGF2_GIVEECCFR
48 и 103
0,008
0,613
ITIH3_ALDLSLK_vs_LYAM1_SYYWIGIR
111 и 120
0,008
0,615
B2MG_VEHSDLSFSK_vs_IGF2_GIVEECCFR
50 и 103
0,016
0,603
ITIH3_ALDLSLK_vs_SHBG_IALGGLLFPASNLR
111 и 18
0,004
0,623
B2MG_VNHVTLSQPK_vs_IGF2_GIVEECCFR
51 и 103
0,008
0,614
KNG1_DIPTNSPELEETLTHTITK_vs_LYAM1_SYYWIGIR
116 и 120
0,013
0,606
B2MG_VNHVTLSQPK_vs_LYAM1_SYYWIGIR
51 и 120
0,016
0,603
KNG1_QVVAGLNFR_vs_IGF2_GIVEECCFR
117 и 103
0,014
0,605
BGH3_LTLLAPLNSVFK_vs_CHL1_VIAVNEVGR
52 и 66
0,017
0,602
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,013
0,607
BGH3_LTLLAPLNSVFK_vs_CRIS3_AVSPPAR
52 и 78
0,017
0,603
LBP_ITGFLKPGK_vs_CRIS3_AVSPPAR
118 и 78
0,019
0,600
BGH3_LTLLAPLNSVFK_vs_CRIS3_YEDLYSNCK
52 и 79
0,007
0,616
LBP_ITGFLKPGK_vs_CRIS3_YEDLYSNCK
118 и 79
0,012
0,608
BGH3_LTLLAPLNSVFK_vs_IGF2_GIVEECCFR
52 и 103
0,013
0,607
LBP_ITGFLKPGK_vs_LYAM1_SYYWIGIR
118 и 120
0,011
0,609
BGH3_LTLLAPLNSVFK_vs_LYAM1_SYYWIGIR
52 и 120
0,008
0,614
LBP_ITGFLKPGK_vs_PSG3_VSAPSGTGHLPGLNPL
118 и 134
0,019
0,600
BGH3_LTLLAPLNSVFK_vs_SHBG_IALGGLLFPASNLR
52 и 18
0,019
0,601
LBP_ITGFLKPGK_vs_SHBG_IALGGLLFPASNLR
118 и 18
0,015
0,604
BGH3_LTLLAPLNSVFK_vs_TENX_LNWEAPPGAFDSFLLR
52 и 141
0,013
0,606
LBP_ITLPDFTGDLR_vs_C163A_INPASLDK
119 и 54
0,011
0,609
C1QB_VPGLYYFTYHASSR_vs_CRIS3_YEDLYSNCK
55 и 79
0,018
0,602
LBP_ITLPDFTGDLR_vs_CRIS3_AVSPPAR
119 и 78
0,004
0,624
C1QB_VPGLYYFTYHASSR_vs_IBP3_FLNVLSPR
55 и 99
0,012
0,608
LBP_ITLPDFTGDLR_vs_CRIS3_YEDLYSNCK
119 и 79
0,002
0,630
C1QB_VPGLYYFTYHASSR_vs_IBP3_YGQPLPGYTTK
55 и 100
0,010
0,610
LBP_ITLPDFTGDLR_vs_IBP3_YGQPLPGYTTK
119 и 100
0,013
0,606
C1QB_VPGLYYFTYHASSR_vs_IGF2_GIVEECCFR
55 и 103
0,002
0,630
LBP_ITLPDFTGDLR_vs_IGF2_GIVEECCFR
119 и 103
0,005
0,621
C1QB_VPGLYYFTYHASSR_vs_LYAM1_SYYWIGIR
55 и 120
0,010
0,611
LBP_ITLPDFTGDLR_vs_LYAM1_SYYWIGIR
119 и 120
0,002
0,635
C1QB_VPGLYYFTYHASSR_vs_PGRP2_AGLLRPDYALLGHR
55 и 126
0,006
0,618
LBP_ITLPDFTGDLR_vs_PGRP2_AGLLRPDYALLGHR
119 и 126
0,004
0,622
C1QB_VPGLYYFTYHASSR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
55 и 135
0,015
0,605
LBP_ITLPDFTGDLR_vs_PSG3_VSAPSGTGHLPGLNPL
119 и 134
0,006
0,617
C1QB_VPGLYYFTYHASSR_vs_SHBG_IALGGLLFPASNLR
55 и 18
0,006
0,617
LBP_ITLPDFTGDLR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
119 и 135
0,017
0,603
C1QB_VPGLYYFTYHASSR_vs_TENX_LNWEAPPGAFDSFLLR
55 и 141
0,013
0,607
LBP_ITLPDFTGDLR_vs_SHBG_IALGGLLFPASNLR
119 и 18
0,004
0,624
CATD_VSTLPAITLK_vs_IGF2_GIVEECCFR
58 и 103
0,018
0,601
LBP_ITLPDFTGDLR_vs_SOM2.CSH_SVEGSCGF
119 и 139
0,015
0,605
CBPN_EALIQFLEQVHQGIK_vs_LYAM1_SYYWIGIR
59 и 120
0,007
0,615
LBP_ITLPDFTGDLR_vs_TENX_LNWEAPPGAFDSFLLR
119 и 141
0,017
0,603
CBPN_EALIQFLEQVHQGIK_vs_SHBG_IALGGLLFPASNLR
59 и 18
0,013
0,606
LBP_ITLPDFTGDLR_vs_VTDB_ELPEHTVK
119 и 147
0,012
0,608
CBPN_NNANGVDLNR_vs_IGF2_GIVEECCFR
60 и 103
0,015
0,604
PEDF_LQSLFDSPDFSK_vs_CRIS3_AVSPPAR
124 и 78
0,007
0,616
CBPN_NNANGVDLNR_vs_LYAM1_SYYWIGIR
60 и 120
0,014
0,606
PEDF_LQSLFDSPDFSK_vs_CRIS3_YEDLYSNCK
124 и 79
0,004
0,623
CD14_LTVGAAQVPAQLLVGALR_vs_IGF2_GIVEECCFR
61 и 103
0,017
0,603
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,005
0,621
CD14_LTVGAAQVPAQLLVGALR_vs_LYAM1_SYYWIGIR
61 и 120
0,012
0,607
PEDF_LQSLFDSPDFSK_vs_TENX_LNWEAPPGAFDSFLLR
124 и 141
0,011
0,610
CD14_SWLAELQQWLKPGLK_vs_LYAM1_SYYWIGIR
62 и 120
0,015
0,605
PEDF_TVQAVLTVPK_vs_CRIS3_YEDLYSNCK
125 и 79
0,010
0,610
CFAB_YGLVTYATYPK_vs_CHL1_VIAVNEVGR
64 и 66
0,013
0,606
PEDF_TVQAVLTVPK_vs_IGF2_GIVEECCFR
125 и 103
0,018
0,602
CFAB_YGLVTYATYPK_vs_CRIS3_AVSPPAR
64 и 78
0,018
0,601
PEDF_TVQAVLTVPK_vs_LYAM1_SYYWIGIR
125 и 120
0,009
0,612
CFAB_YGLVTYATYPK_vs_CRIS3_YEDLYSNCK
64 и 79
0,010
0,611
PEDF_TVQAVLTVPK_vs_TENX_LNWEAPPGAFDSFLLR
125 и 141
0,016
0,603
CFAB_YGLVTYATYPK_vs_IBP3_FLNVLSPR
64 и 99
0,016
0,603
PSG2_IHPSYTNYR_vs_CRIS3_YEDLYSNCK
133 и 79
0,018
0,602
CFAB_YGLVTYATYPK_vs_IBP3_YGQPLPGYTTK
64 и 100
0,010
0,610
PSG2_IHPSYTNYR_vs_LYAM1_SYYWIGIR
133 и 120
0,015
0,604
CFAB_YGLVTYATYPK_vs_IGF2_GIVEECCFR
64 и 103
0,003
0,628
PSG2_IHPSYTNYR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
133 и 135
0,014
0,605
CFAB_YGLVTYATYPK_vs_LYAM1_SYYWIGIR
64 и 120
0,004
0,623
VTNC_GQYCYELDEK_vs_ALS_IRPHTFTGLSGLR
149 и 40
0,013
0,607
CFAB_YGLVTYATYPK_vs_PGRP2_AGLLRPDYALLGHR
64 и 126
0,012
0,608
VTNC_GQYCYELDEK_vs_CHL1_VIAVNEVGR
149 и 66
0,005
0,621
CFAB_YGLVTYATYPK_vs_PSG3_VSAPSGTGHLPGLNPL
64 и 134
0,006
0,619
VTNC_GQYCYELDEK_vs_CRIS3_AVSPPAR
149 и 78
0,005
0,621
CFAB_YGLVTYATYPK_vs_SHBG_IALGGLLFPASNLR
64 и 18
0,009
0,613
VTNC_GQYCYELDEK_vs_CRIS3_YEDLYSNCK
149 и 79
0,003
0,627
CFAB_YGLVTYATYPK_vs_TENX_LNWEAPPGAFDSFLLR
64 и 141
0,019
0,600
VTNC_GQYCYELDEK_vs_IBP3_FLNVLSPR
149 и 99
0,002
0,630
CFAB_YGLVTYATYPK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
64 и 144
0,005
0,620
VTNC_GQYCYELDEK_vs_IBP3_YGQPLPGYTTK
149 и 100
0,001
0,647
CLUS_ASSIIDELFQDR_vs_CRIS3_YEDLYSNCK
67 и 79
0,019
0,601
VTNC_GQYCYELDEK_vs_IGF2_GIVEECCFR
149 и 103
0,000
0,653
CLUS_ASSIIDELFQDR_vs_IGF2_GIVEECCFR
67 и 103
0,014
0,605
VTNC_GQYCYELDEK_vs_ITIH4_ILDDLSPR
149 и 112
0,011
0,609
CLUS_ASSIIDELFQDR_vs_LYAM1_SYYWIGIR
67 и 120
0,016
0,603
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,000
0,650
CO5_TLLPVSKPEIR_vs_CRIS3_AVSPPAR
70 и 78
0,015
0,604
VTNC_GQYCYELDEK_vs_NCAM1_GLGEISAASEFK
149 и 121
0,015
0,604
CO5_TLLPVSKPEIR_vs_CRIS3_YEDLYSNCK
70 и 79
0,009
0,612
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,002
0,635
CO5_TLLPVSKPEIR_vs_IGF2_GIVEECCFR
70 и 103
0,008
0,614
VTNC_GQYCYELDEK_vs_PSG3_VSAPSGTGHLPGLNPL
149 и 134
0,001
0,640
CO5_TLLPVSKPEIR_vs_LYAM1_SYYWIGIR
70 и 120
0,004
0,625
VTNC_GQYCYELDEK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
149 и 135
0,011
0,609
CO5_TLLPVSKPEIR_vs_PGRP2_AGLLRPDYALLGHR
70 и 126
0,009
0,611
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,001
0,640
CO5_TLLPVSKPEIR_vs_PSG3_VSAPSGTGHLPGLNPL
70 и 134
0,007
0,616
VTNC_GQYCYELDEK_vs_SOM2.CSH_SVEGSCGF
149 и 139
0,020
0,600
CO5_TLLPVSKPEIR_vs_SHBG_IALGGLLFPASNLR
70 и 18
0,008
0,613
VTNC_GQYCYELDEK_vs_SPRL1_VLTHSELAPLR
149 и 140
0,013
0,607
CO5_TLLPVSKPEIR_vs_TENX_LNWEAPPGAFDSFLLR
70 и 141
0,019
0,601
VTNC_GQYCYELDEK_vs_TENX_LNWEAPPGAFDSFLLR
149 и 141
0,005
0,620
CO5_VFQFLEK_vs_CRIS3_AVSPPAR
71 и 78
0,019
0,601
VTNC_GQYCYELDEK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
149 и 144
0,004
0,624
CO5_VFQFLEK_vs_CRIS3_YEDLYSNCK
71 и 79
0,013
0,607
VTNC_GQYCYELDEK_vs_VTDB_ELPEHTVK
149 и 147
0,001
0,646
CO5_VFQFLEK_vs_IGF2_GIVEECCFR
71 и 103
0,014
0,606
VTNC_VDTVDPPYPR_vs_CHL1_VIAVNEVGR
150 и 66
0,007
0,616
CO5_VFQFLEK_vs_LYAM1_SYYWIGIR
71 и 120
0,005
0,619
VTNC_VDTVDPPYPR_vs_CRIS3_AVSPPAR
150 и 78
0,004
0,624
CO5_VFQFLEK_vs_PGRP2_AGLLRPDYALLGHR
71 и 126
0,013
0,607
VTNC_VDTVDPPYPR_vs_CRIS3_YEDLYSNCK
150 и 79
0,003
0,629
CO5_VFQFLEK_vs_PSG3_VSAPSGTGHLPGLNPL
71 и 134
0,016
0,604
VTNC_VDTVDPPYPR_vs_IBP3_FLNVLSPR
150 и 99
0,006
0,618
CO5_VFQFLEK_vs_SHBG_IALGGLLFPASNLR
71 и 18
0,010
0,610
VTNC_VDTVDPPYPR_vs_IBP3_YGQPLPGYTTK
150 и 100
0,002
0,630
CO5_VFQFLEK_vs_TENX_LNWEAPPGAFDSFLLR
71 и 141
0,019
0,601
VTNC_VDTVDPPYPR_vs_IGF2_GIVEECCFR
150 и 103
0,001
0,641
CO6_ALNHLPLEYNSALYSR_vs_CHL1_VIAVNEVGR
72 и 66
0,017
0,602
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,000
0,653
CO6_ALNHLPLEYNSALYSR_vs_IGF2_GIVEECCFR
72 и 103
0,009
0,612
VTNC_VDTVDPPYPR_vs_NCAM1_GLGEISAASEFK
150 и 121
0,013
0,607
CO6_ALNHLPLEYNSALYSR_vs_LYAM1_SYYWIGIR
72 и 120
0,011
0,609
VTNC_VDTVDPPYPR_vs_PGRP2_AGLLRPDYALLGHR
150 и 126
0,004
0,624
CO6_ALNHLPLEYNSALYSR_vs_PGRP2_AGLLRPDYALLGHR
72 и 126
0,017
0,602
VTNC_VDTVDPPYPR_vs_PSG3_VSAPSGTGHLPGLNPL
150 и 134
0,002
0,630
CO8A_SLLQPNK_vs_IGF2_GIVEECCFR
74 и 103
0,008
0,613
VTNC_VDTVDPPYPR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
150 и 135
0,012
0,608
CO8A_SLLQPNK_vs_LYAM1_SYYWIGIR
74 и 120
0,018
0,602
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,002
0,633
CO8A_SLLQPNK_vs_TENX_LNWEAPPGAFDSFLLR
74 и 141
0,016
0,603
VTNC_VDTVDPPYPR_vs_SPRL1_VLTHSELAPLR
150 и 140
0,018
0,601
CO8B_QALEEFQK_vs_IGF2_GIVEECCFR
76 и 103
0,006
0,617
VTNC_VDTVDPPYPR_vs_TENX_LNWEAPPGAFDSFLLR
150 и 141
0,009
0,612
CO8B_QALEEFQK_vs_TENX_LNWEAPPGAFDSFLLR
76 и 141
0,010
0,610
VTNC_VDTVDPPYPR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
150 и 144
0,012
0,608
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_IGF2_GIVEECCFR
82 и 103
0,018
0,601
VTNC_VDTVDPPYPR_vs_VTDB_ELPEHTVK
150 и 147
0,001
0,648
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_LYAM1_SYYWIGIR
82 и 120
0,005
0,620
Таблица 45: эффективность классификации по обращениям, недели с 17 до 21.
AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, без BMI-стратификации.
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
A2GL_DLLLPQPDLR_vs_IBP1_VVESLAK
34 и 97
0,046
0,615
CFAB_YGLVTYATYPK_vs_LYAM1_SYYWIGIR
64 и 120
0,044
0,616
A2GL_DLLLPQPDLR_vs_LYAM1_SYYWIGIR
34 и 120
0,023
0,631
CLUS_ASSIIDELFQDR_vs_CRIS3_AVSPPAR
67 и 78
0,028
0,627
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,013
0,642
CLUS_ASSIIDELFQDR_vs_CRIS3_YEDLYSNCK
67 и 79
0,016
0,638
AFAM_DADPDTFFAK_vs_LYAM1_SYYWIGIR
37 и 120
0,012
0,644
CLUS_ASSIIDELFQDR_vs_IBP1_VVESLAK
67 и 97
0,039
0,619
AFAM_DADPDTFFAK_vs_PGRP2_AGLLRPDYALLGHR
37 и 126
0,041
0,618
CLUS_ASSIIDELFQDR_vs_IBP3_YGQPLPGYTTK
67 и 100
0,047
0,614
AFAM_DADPDTFFAK_vs_SHBG_IALGGLLFPASNLR
37 и 18
0,050
0,613
CLUS_ASSIIDELFQDR_vs_IGF2_GIVEECCFR
67 и 103
0,011
0,645
AFAM_HFQNLGK_vs_ALS_IRPHTFTGLSGLR
38 и 40
0,035
0,621
CLUS_ASSIIDELFQDR_vs_LYAM1_SYYWIGIR
67 и 120
0,001
0,683
AFAM_HFQNLGK_vs_CRIS3_YEDLYSNCK
38 и 79
0,036
0,621
CLUS_ASSIIDELFQDR_vs_PGRP2_AGLLRPDYALLGHR
67 и 126
0,026
0,628
AFAM_HFQNLGK_vs_IBP1_VVESLAK
38 и 97
0,044
0,618
CLUS_ASSIIDELFQDR_vs_PSG3_VSAPSGTGHLPGLNPL
67 и 134
0,037
0,620
AFAM_HFQNLGK_vs_IBP3_FLNVLSPR
38 и 99
0,048
0,614
CLUS_ASSIIDELFQDR_vs_SHBG_IALGGLLFPASNLR
67 и 18
0,017
0,638
AFAM_HFQNLGK_vs_IBP3_YGQPLPGYTTK
38 и 100
0,013
0,642
CLUS_ASSIIDELFQDR_vs_VTDB_ELPEHTVK
67 и 147
0,013
0,643
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,005
0,661
CLUS_LFDSDPITVTVPVEVSR_vs_CRIS3_AVSPPAR
68 и 78
0,028
0,627
AFAM_HFQNLGK_vs_LYAM1_SYYWIGIR
38 и 120
0,010
0,648
CLUS_LFDSDPITVTVPVEVSR_vs_CRIS3_YEDLYSNCK
68 и 79
0,014
0,642
AFAM_HFQNLGK_vs_PGRP2_AGLLRPDYALLGHR
38 и 126
0,041
0,617
CLUS_LFDSDPITVTVPVEVSR_vs_IBP1_VVESLAK
68 и 97
0,039
0,619
ANGT_DPTFIPAPIQAK_vs_LYAM1_SYYWIGIR
42 и 120
0,010
0,648
CLUS_LFDSDPITVTVPVEVSR_vs_IBP3_YGQPLPGYTTK
68 и 100
0,046
0,615
ANGT_DPTFIPAPIQAK_vs_SHBG_IALGGLLFPASNLR
42 и 18
0,027
0,627
CLUS_LFDSDPITVTVPVEVSR_vs_IGF2_GIVEECCFR
68 и 103
0,015
0,640
APOC3_GWVTDGFSSLK_vs_CRIS3_YEDLYSNCK
47 и 79
0,042
0,617
CLUS_LFDSDPITVTVPVEVSR_vs_LYAM1_SYYWIGIR
68 и 120
0,004
0,668
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,015
0,639
CLUS_LFDSDPITVTVPVEVSR_vs_PGRP2_AGLLRPDYALLGHR
68 и 126
0,049
0,613
APOH_ATVVYQGER_vs_CRIS3_YEDLYSNCK
48 и 79
0,027
0,627
CLUS_LFDSDPITVTVPVEVSR_vs_SHBG_IALGGLLFPASNLR
68 и 18
0,023
0,631
APOH_ATVVYQGER_vs_IBP1_VVESLAK
48 и 97
0,046
0,615
CLUS_LFDSDPITVTVPVEVSR_vs_VTDB_ELPEHTVK
68 и 147
0,014
0,642
APOH_ATVVYQGER_vs_LYAM1_SYYWIGIR
48 и 120
0,011
0,647
CO5_TLLPVSKPEIR_vs_CRIS3_YEDLYSNCK
70 и 79
0,036
0,620
APOH_ATVVYQGER_vs_PGRP2_AGLLRPDYALLGHR
48 и 126
0,045
0,615
CO5_TLLPVSKPEIR_vs_LYAM1_SYYWIGIR
70 и 120
0,005
0,663
B2MG_VEHSDLSFSK_vs_LYAM1_SYYWIGIR
50 и 120
0,009
0,651
CO5_TLLPVSKPEIR_vs_SHBG_IALGGLLFPASNLR
70 и 18
0,043
0,616
B2MG_VNHVTLSQPK_vs_CRIS3_AVSPPAR
51 и 78
0,040
0,618
CO5_VFQFLEK_vs_LYAM1_SYYWIGIR
71 и 120
0,015
0,641
B2MG_VNHVTLSQPK_vs_CRIS3_YEDLYSNCK
51 и 79
0,024
0,630
CO6_ALNHLPLEYNSALYSR_vs_LYAM1_SYYWIGIR
72 и 120
0,006
0,657
B2MG_VNHVTLSQPK_vs_IBP1_VVESLAK
51 и 97
0,047
0,614
CO6_ALNHLPLEYNSALYSR_vs_SHBG_IALGGLLFPASNLR
72 и 18
0,039
0,619
B2MG_VNHVTLSQPK_vs_IGF2_GIVEECCFR
51 и 103
0,040
0,618
CO8A_SLLQPNK_vs_IBP1_VVESLAK
74 и 97
0,046
0,615
B2MG_VNHVTLSQPK_vs_LYAM1_SYYWIGIR
51 и 120
0,002
0,676
CO8A_SLLQPNK_vs_IGF2_GIVEECCFR
74 и 103
0,047
0,614
B2MG_VNHVTLSQPK_vs_SHBG_IALGGLLFPASNLR
51 и 18
0,032
0,624
CO8A_SLLQPNK_vs_LYAM1_SYYWIGIR
74 и 120
0,007
0,654
CATD_VGFAEAAR_vs_ALS_IRPHTFTGLSGLR
57 и 40
0,022
0,632
CO8B_QALEEFQK_vs_IBP1_VVESLAK
76 и 97
0,036
0,621
CATD_VGFAEAAR_vs_C163A_INPASLDK
57 и 54
0,001
0,697
CO8B_QALEEFQK_vs_IGF2_GIVEECCFR
76 и 103
0,046
0,615
CATD_VGFAEAAR_vs_CRIS3_AVSPPAR
57 и 78
0,013
0,643
CO8B_QALEEFQK_vs_LYAM1_SYYWIGIR
76 и 120
0,018
0,636
CATD_VGFAEAAR_vs_CRIS3_YEDLYSNCK
57 и 79
0,008
0,653
CO8B_QALEEFQK_vs_PSG3_VSAPSGTGHLPGLNPL
76 и 134
0,046
0,615
CATD_VGFAEAAR_vs_CSH_AHQLAIDTYQEFEETYIPK
57 и 80
0,005
0,661
F13B_GDTYPAELYITGSILR_vs_CRIS3_AVSPPAR
84 и 78
0,021
0,633
CATD_VGFAEAAR_vs_CSH_ISLLLIESWLEPVR
57 и 81
0,009
0,651
F13B_GDTYPAELYITGSILR_vs_CRIS3_YEDLYSNCK
84 и 79
0,013
0,643
CATD_VGFAEAAR_vs_FBLN1_TGYYFDGISR
57 и 86
0,018
0,636
F13B_GDTYPAELYITGSILR_vs_IBP1_VVESLAK
84 и 97
0,033
0,623
CATD_VGFAEAAR_vs_IBP1_VVESLAK
57 и 97
0,010
0,648
F13B_GDTYPAELYITGSILR_vs_IBP3_YGQPLPGYTTK
84 и 100
0,039
0,619
CATD_VGFAEAAR_vs_IBP2_LIQGAPTIR
57 и 98
0,050
0,613
F13B_GDTYPAELYITGSILR_vs_IGF2_GIVEECCFR
84 и 103
0,011
0,646
CATD_VGFAEAAR_vs_IBP3_FLNVLSPR
57 и 99
0,026
0,628
F13B_GDTYPAELYITGSILR_vs_LYAM1_SYYWIGIR
84 и 120
0,001
0,695
CATD_VGFAEAAR_vs_IBP3_YGQPLPGYTTK
57 и 100
0,019
0,635
F13B_GDTYPAELYITGSILR_vs_PGRP2_AGLLRPDYALLGHR
84 и 126
0,031
0,624
CATD_VGFAEAAR_vs_IGF2_GIVEECCFR
57 и 103
0,003
0,669
F13B_GDTYPAELYITGSILR_vs_SHBG_IALGGLLFPASNLR
84 и 18
0,020
0,634
CATD_VGFAEAAR_vs_ITIH4_ILDDLSPR
57 и 112
0,020
0,634
F13B_GDTYPAELYITGSILR_vs_SPRL1_VLTHSELAPLR
84 и 140
0,031
0,624
CATD_VGFAEAAR_vs_LYAM1_SYYWIGIR
57 и 120
0,001
0,688
FETUA_FSVVYAK_vs_CRIS3_YEDLYSNCK
88 и 79
0,045
0,615
CATD_VGFAEAAR_vs_PGRP2_AGLLRPDYALLGHR
57 и 126
0,009
0,650
FETUA_FSVVYAK_vs_LYAM1_SYYWIGIR
88 и 120
0,007
0,654
CATD_VGFAEAAR_vs_PSG3_VSAPSGTGHLPGLNPL
57 и 134
0,008
0,652
FETUA_HTLNQIDEVK_vs_LYAM1_SYYWIGIR
89 и 120
0,016
0,638
CATD_VGFAEAAR_vs_SHBG_IALGGLLFPASNLR
57 и 18
0,012
0,644
HABP2_FLNWIK_vs_LYAM1_SYYWIGIR
92 и 120
0,033
0,623
CATD_VGFAEAAR_vs_SOM2.CSH_NYGLLYCFR
57 и 138
0,010
0,649
HEMO_NFPSPVDAAFR_vs_LYAM1_SYYWIGIR
93 и 120
0,019
0,635
CATD_VGFAEAAR_vs_SOM2.CSH_SVEGSCGF
57 и 139
0,007
0,655
IBP4_QCHPALDGQR_vs_CRIS3_AVSPPAR
2 и 78
0,021
0,632
CATD_VGFAEAAR_vs_SPRL1_VLTHSELAPLR
57 и 140
0,009
0,650
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,011
0,647
CATD_VGFAEAAR_vs_TENX_LNWEAPPGAFDSFLLR
57 и 141
0,004
0,666
IBP4_QCHPALDGQR_vs_IBP1_VVESLAK
2 и 97
0,042
0,617
CATD_VGFAEAAR_vs_TENX_LSQLSVTDVTTSSLR
57 и 142
0,013
0,643
IBP4_QCHPALDGQR_vs_IGF2_GIVEECCFR
2 и 103
0,047
0,614
CATD_VGFAEAAR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
57 и 144
0,014
0,642
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,003
0,669
CATD_VGFAEAAR_vs_VTDB_ELPEHTVK
57 и 147
0,023
0,631
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,033
0,623
CATD_VSTLPAITLK_vs_ALS_IRPHTFTGLSGLR
58 и 40
0,020
0,634
IBP6_HLDSVLQQLQTEVYR_vs_CRIS3_YEDLYSNCK
102 и 79
0,044
0,616
CATD_VSTLPAITLK_vs_C163A_INPASLDK
58 и 54
0,004
0,664
IBP6_HLDSVLQQLQTEVYR_vs_LYAM1_SYYWIGIR
102 и 120
0,022
0,631
CATD_VSTLPAITLK_vs_CRIS3_AVSPPAR
58 и 78
0,012
0,645
ITIH3_ALDLSLK_vs_SHBG_IALGGLLFPASNLR
111 и 18
0,039
0,619
CATD_VSTLPAITLK_vs_CRIS3_YEDLYSNCK
58 и 79
0,006
0,657
KNG1_DIPTNSPELEETLTHTITK_vs_IBP1_VVESLAK
116 и 97
0,048
0,614
CATD_VSTLPAITLK_vs_CSH_AHQLAIDTYQEFEETYIPK
58 и 80
0,013
0,644
KNG1_DIPTNSPELEETLTHTITK_vs_LYAM1_SYYWIGIR
116 и 120
0,012
0,645
CATD_VSTLPAITLK_vs_CSH_ISLLLIESWLEPVR
58 и 81
0,023
0,631
KNG1_QVVAGLNFR_vs_CRIS3_AVSPPAR
117 и 78
0,041
0,618
CATD_VSTLPAITLK_vs_FBLN1_TGYYFDGISR
58 и 86
0,017
0,638
KNG1_QVVAGLNFR_vs_CRIS3_YEDLYSNCK
117 и 79
0,020
0,634
CATD_VSTLPAITLK_vs_IBP1_VVESLAK
58 и 97
0,012
0,645
KNG1_QVVAGLNFR_vs_IBP1_VVESLAK
117 и 97
0,024
0,630
CATD_VSTLPAITLK_vs_IBP3_FLNVLSPR
58 и 99
0,017
0,637
KNG1_QVVAGLNFR_vs_IGF2_GIVEECCFR
117 и 103
0,020
0,634
CATD_VSTLPAITLK_vs_IBP3_YGQPLPGYTTK
58 и 100
0,013
0,643
KNG1_QVVAGLNFR_vs_ITIH4_ILDDLSPR
117 и 112
0,038
0,619
CATD_VSTLPAITLK_vs_IGF2_GIVEECCFR
58 и 103
0,003
0,670
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,002
0,682
CATD_VSTLPAITLK_vs_ITIH4_ILDDLSPR
58 и 112
0,045
0,615
KNG1_QVVAGLNFR_vs_PGRP2_AGLLRPDYALLGHR
117 и 126
0,027
0,627
CATD_VSTLPAITLK_vs_LYAM1_SYYWIGIR
58 и 120
0,001
0,684
KNG1_QVVAGLNFR_vs_SHBG_IALGGLLFPASNLR
117 и 18
0,016
0,639
CATD_VSTLPAITLK_vs_PGRP2_AGLLRPDYALLGHR
58 и 126
0,010
0,649
KNG1_QVVAGLNFR_vs_VTDB_ELPEHTVK
117 и 147
0,041
0,618
CATD_VSTLPAITLK_vs_PSG3_VSAPSGTGHLPGLNPL
58 и 134
0,006
0,658
LBP_ITLPDFTGDLR_vs_LYAM1_SYYWIGIR
119 и 120
0,031
0,624
CATD_VSTLPAITLK_vs_SHBG_IALGGLLFPASNLR
58 и 18
0,012
0,644
PEDF_LQSLFDSPDFSK_vs_CRIS3_AVSPPAR
124 и 78
0,034
0,622
CATD_VSTLPAITLK_vs_SOM2.CSH_NYGLLYCFR
58 и 138
0,023
0,631
PEDF_LQSLFDSPDFSK_vs_CRIS3_YEDLYSNCK
124 и 79
0,026
0,628
CATD_VSTLPAITLK_vs_SOM2.CSH_SVEGSCGF
58 и 139
0,020
0,634
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,004
0,664
CATD_VSTLPAITLK_vs_SPRL1_VLTHSELAPLR
58 и 140
0,015
0,640
PEDF_TVQAVLTVPK_vs_CRIS3_AVSPPAR
125 и 78
0,027
0,627
CATD_VSTLPAITLK_vs_TENX_LNWEAPPGAFDSFLLR
58 и 141
0,004
0,666
PEDF_TVQAVLTVPK_vs_CRIS3_YEDLYSNCK
125 и 79
0,017
0,638
CATD_VSTLPAITLK_vs_TENX_LSQLSVTDVTTSSLR
58 и 142
0,015
0,640
PEDF_TVQAVLTVPK_vs_IGF2_GIVEECCFR
125 и 103
0,029
0,626
CATD_VSTLPAITLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
58 и 144
0,026
0,628
PEDF_TVQAVLTVPK_vs_LYAM1_SYYWIGIR
125 и 120
0,002
0,677
CATD_VSTLPAITLK_vs_VTDB_ELPEHTVK
58 и 147
0,037
0,620
PSG2_IHPSYTNYR_vs_CRIS3_AVSPPAR
133 и 78
0,015
0,640
CBPN_EALIQFLEQVHQGIK_vs_LYAM1_SYYWIGIR
59 и 120
0,020
0,634
PSG2_IHPSYTNYR_vs_CRIS3_YEDLYSNCK
133 и 79
0,011
0,646
CBPN_NNANGVDLNR_vs_LYAM1_SYYWIGIR
60 и 120
0,007
0,656
PSG2_IHPSYTNYR_vs_FBLN1_TGYYFDGISR
133 и 86
0,020
0,634
CBPN_NNANGVDLNR_vs_SPRL1_VLTHSELAPLR
60 и 140
0,026
0,628
PSG2_IHPSYTNYR_vs_IBP1_VVESLAK
133 и 97
0,027
0,627
CD14_LTVGAAQVPAQLLVGALR_vs_CRIS3_YEDLYSNCK
61 и 79
0,028
0,627
PSG2_IHPSYTNYR_vs_IBP2_LIQGAPTIR
133 и 98
0,028
0,626
CD14_LTVGAAQVPAQLLVGALR_vs_CSH_AHQLAIDTYQEFEETYIPK
61 и 80
0,044
0,616
PSG2_IHPSYTNYR_vs_IGF2_GIVEECCFR
133 и 103
0,042
0,617
CD14_LTVGAAQVPAQLLVGALR_vs_IBP1_VVESLAK
61 и 97
0,019
0,635
PSG2_IHPSYTNYR_vs_LYAM1_SYYWIGIR
133 и 120
0,006
0,658
CD14_LTVGAAQVPAQLLVGALR_vs_IGF2_GIVEECCFR
61 и 103
0,015
0,640
PSG2_IHPSYTNYR_vs_PSG3_VSAPSGTGHLPGLNPL
133 и 134
0,025
0,629
CD14_LTVGAAQVPAQLLVGALR_vs_LYAM1_SYYWIGIR
61 и 120
0,002
0,681
PSG2_IHPSYTNYR_vs_SHBG_IALGGLLFPASNLR
133 и 18
0,014
0,642
CD14_LTVGAAQVPAQLLVGALR_vs_PGRP2_AGLLRPDYALLGHR
61 и 126
0,009
0,650
PSG2_IHPSYTNYR_vs_SPRL1_VLTHSELAPLR
133 и 140
0,036
0,621
CD14_LTVGAAQVPAQLLVGALR_vs_PSG3_VSAPSGTGHLPGLNPL
61 и 134
0,045
0,615
PTGDS_GPGEDFR_vs_CRIS3_YEDLYSNCK
137 и 79
0,036
0,621
CD14_LTVGAAQVPAQLLVGALR_vs_SHBG_IALGGLLFPASNLR
61 и 18
0,011
0,647
PTGDS_GPGEDFR_vs_IBP1_VVESLAK
137 и 97
0,050
0,613
CD14_LTVGAAQVPAQLLVGALR_vs_SOM2.CSH_SVEGSCGF
61 и 139
0,033
0,623
PTGDS_GPGEDFR_vs_LYAM1_SYYWIGIR
137 и 120
0,018
0,636
CD14_LTVGAAQVPAQLLVGALR_vs_SPRL1_VLTHSELAPLR
61 и 140
0,034
0,622
THBG_AVLHIGEK_vs_LYAM1_SYYWIGIR
143 и 120
0,015
0,640
CD14_LTVGAAQVPAQLLVGALR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
61 и 144
0,020
0,633
VTNC_GQYCYELDEK_vs_CRIS3_YEDLYSNCK
149 и 79
0,033
0,622
CD14_LTVGAAQVPAQLLVGALR_vs_VTDB_ELPEHTVK
61 и 147
0,011
0,647
VTNC_GQYCYELDEK_vs_IBP1_VVESLAK
149 и 97
0,048
0,614
CD14_SWLAELQQWLKPGLK_vs_CRIS3_YEDLYSNCK
62 и 79
0,033
0,623
VTNC_GQYCYELDEK_vs_IGF2_GIVEECCFR
149 и 103
0,029
0,625
CD14_SWLAELQQWLKPGLK_vs_IBP1_VVESLAK
62 и 97
0,021
0,633
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,002
0,675
CD14_SWLAELQQWLKPGLK_vs_IGF2_GIVEECCFR
62 и 103
0,025
0,629
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,044
0,616
CD14_SWLAELQQWLKPGLK_vs_LYAM1_SYYWIGIR
62 и 120
0,003
0,669
VTNC_GQYCYELDEK_vs_PSG3_VSAPSGTGHLPGLNPL
149 и 134
0,032
0,623
CD14_SWLAELQQWLKPGLK_vs_PGRP2_AGLLRPDYALLGHR
62 и 126
0,015
0,640
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,030
0,625
CD14_SWLAELQQWLKPGLK_vs_SHBG_IALGGLLFPASNLR
62 и 18
0,017
0,638
VTNC_VDTVDPPYPR_vs_CRIS3_YEDLYSNCK
150 и 79
0,040
0,618
CD14_SWLAELQQWLKPGLK_vs_SOM2.CSH_SVEGSCGF
62 и 139
0,041
0,618
VTNC_VDTVDPPYPR_vs_IGF2_GIVEECCFR
150 и 103
0,049
0,613
CD14_SWLAELQQWLKPGLK_vs_SPRL1_VLTHSELAPLR
62 и 140
0,049
0,613
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,004
0,665
CD14_SWLAELQQWLKPGLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
62 и 144
0,024
0,630
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,038
0,619
CD14_SWLAELQQWLKPGLK_vs_VTDB_ELPEHTVK
62 и 147
0,014
0,641
Таблица 46: эффективность классификации по обращениям, недели с 17 до 21.
AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, с BMI-стратификацией (>22≤37).
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
A2GL_DLLLPQPDLR_vs_CRIS3_YEDLYSNCK
34 и 79
0,047
0,637
CLUS_LFDSDPITVTVPVEVSR_vs_CRIS3_YEDLYSNCK
68 и 79
0,027
0,652
A2GL_DLLLPQPDLR_vs_LYAM1_SYYWIGIR
34 и 120
0,006
0,689
CLUS_LFDSDPITVTVPVEVSR_vs_LYAM1_SYYWIGIR
68 и 120
0,021
0,659
AFAM_DADPDTFFAK_vs_CRIS3_YEDLYSNCK
37 и 79
0,027
0,652
CO5_TLLPVSKPEIR_vs_CRIS3_YEDLYSNCK
70 и 79
0,047
0,637
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,018
0,663
CO5_TLLPVSKPEIR_vs_LYAM1_SYYWIGIR
70 и 120
0,006
0,688
AFAM_DADPDTFFAK_vs_LYAM1_SYYWIGIR
37 и 120
0,007
0,687
CO5_VFQFLEK_vs_LYAM1_SYYWIGIR
71 и 120
0,024
0,655
AFAM_DADPDTFFAK_vs_PGRP2_AGLLRPDYALLGHR
37 и 126
0,016
0,666
CO6_ALNHLPLEYNSALYSR_vs_LYAM1_SYYWIGIR
72 и 120
0,006
0,689
AFAM_DADPDTFFAK_vs_SOM2.CSH_SVEGSCGF
37 и 139
0,049
0,636
CO6_ALNHLPLEYNSALYSR_vs_PGRP2_AGLLRPDYALLGHR
72 и 126
0,026
0,653
AFAM_HFQNLGK_vs_ALS_IRPHTFTGLSGLR
38 и 40
0,035
0,645
CO8A_SLLQPNK_vs_LYAM1_SYYWIGIR
74 и 120
0,012
0,674
AFAM_HFQNLGK_vs_CRIS3_AVSPPAR
38 и 78
0,043
0,640
CO8B_QALEEFQK_vs_LYAM1_SYYWIGIR
76 и 120
0,036
0,644
AFAM_HFQNLGK_vs_CRIS3_YEDLYSNCK
38 и 79
0,020
0,660
F13B_GDTYPAELYITGSILR_vs_CRIS3_AVSPPAR
84 и 78
0,016
0,665
AFAM_HFQNLGK_vs_IBP3_FLNVLSPR
38 и 99
0,039
0,642
F13B_GDTYPAELYITGSILR_vs_CRIS3_YEDLYSNCK
84 и 79
0,012
0,672
AFAM_HFQNLGK_vs_IBP3_YGQPLPGYTTK
38 и 100
0,031
0,648
F13B_GDTYPAELYITGSILR_vs_IGF2_GIVEECCFR
84 и 103
0,035
0,646
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,009
0,680
F13B_GDTYPAELYITGSILR_vs_LYAM1_SYYWIGIR
84 и 120
0,001
0,729
AFAM_HFQNLGK_vs_LYAM1_SYYWIGIR
38 и 120
0,005
0,693
F13B_GDTYPAELYITGSILR_vs_PGRP2_AGLLRPDYALLGHR
84 и 126
0,015
0,668
AFAM_HFQNLGK_vs_PGRP2_AGLLRPDYALLGHR
38 и 126
0,013
0,672
F13B_GDTYPAELYITGSILR_vs_SHBG_IALGGLLFPASNLR
84 и 18
0,031
0,649
AFAM_HFQNLGK_vs_SOM2.CSH_SVEGSCGF
38 и 139
0,026
0,653
FETUA_FSVVYAK_vs_LYAM1_SYYWIGIR
88 и 120
0,013
0,671
ANGT_DPTFIPAPIQAK_vs_CRIS3_AVSPPAR
42 и 78
0,035
0,645
FETUA_HTLNQIDEVK_vs_LYAM1_SYYWIGIR
89 и 120
0,011
0,674
ANGT_DPTFIPAPIQAK_vs_CRIS3_YEDLYSNCK
42 и 79
0,031
0,648
HEMO_NFPSPVDAAFR_vs_LYAM1_SYYWIGIR
93 и 120
0,026
0,653
ANGT_DPTFIPAPIQAK_vs_LYAM1_SYYWIGIR
42 и 120
0,004
0,697
IBP4_QCHPALDGQR_vs_CRIS3_AVSPPAR
2 и 78
0,035
0,645
ANGT_DPTFIPAPIQAK_vs_PGRP2_AGLLRPDYALLGHR
42 и 126
0,029
0,650
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,017
0,664
ANGT_DPTFIPAPIQAK_vs_SHBG_IALGGLLFPASNLR
42 и 18
0,028
0,651
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,003
0,706
ANGT_DPTFIPAPIQAK_vs_SOM2.CSH_SVEGSCGF
42 и 139
0,049
0,636
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,046
0,638
APOH_ATVVYQGER_vs_CRIS3_YEDLYSNCK
48 и 79
0,029
0,650
IBP6_HLDSVLQQLQTEVYR_vs_LYAM1_SYYWIGIR
102 и 120
0,017
0,664
APOH_ATVVYQGER_vs_LYAM1_SYYWIGIR
48 и 120
0,016
0,667
ITIH3_ALDLSLK_vs_LYAM1_SYYWIGIR
111 и 120
0,028
0,652
APOH_ATVVYQGER_vs_PGRP2_AGLLRPDYALLGHR
48 и 126
0,016
0,665
ITIH3_ALDLSLK_vs_SHBG_IALGGLLFPASNLR
111 и 18
0,049
0,636
B2MG_VEHSDLSFSK_vs_LYAM1_SYYWIGIR
50 и 120
0,015
0,668
KNG1_DIPTNSPELEETLTHTITK_vs_LYAM1_SYYWIGIR
116 и 120
0,011
0,675
B2MG_VNHVTLSQPK_vs_CRIS3_AVSPPAR
51 и 78
0,044
0,639
KNG1_QVVAGLNFR_vs_CRIS3_AVSPPAR
117 и 78
0,034
0,646
B2MG_VNHVTLSQPK_vs_CRIS3_YEDLYSNCK
51 и 79
0,031
0,649
KNG1_QVVAGLNFR_vs_CRIS3_YEDLYSNCK
117 и 79
0,020
0,661
B2MG_VNHVTLSQPK_vs_LYAM1_SYYWIGIR
51 и 120
0,002
0,709
KNG1_QVVAGLNFR_vs_IGF2_GIVEECCFR
117 и 103
0,035
0,645
CATD_VGFAEAAR_vs_ALS_IRPHTFTGLSGLR
57 и 40
0,031
0,649
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,002
0,714
CATD_VGFAEAAR_vs_C163A_INPASLDK
57 и 54
0,001
0,722
KNG1_QVVAGLNFR_vs_PGRP2_AGLLRPDYALLGHR
117 и 126
0,025
0,654
CATD_VGFAEAAR_vs_CRIS3_AVSPPAR
57 и 78
0,018
0,664
PAPP1_DIPHWLNPTR_vs_C163A_INPASLDK
122 и 54
0,050
0,635
CATD_VGFAEAAR_vs_CRIS3_YEDLYSNCK
57 и 79
0,010
0,677
PAPP1_DIPHWLNPTR_vs_CRIS3_AVSPPAR
122 и 78
0,014
0,669
CATD_VGFAEAAR_vs_CSH_AHQLAIDTYQEFEETYIPK
57 и 80
0,008
0,682
PAPP1_DIPHWLNPTR_vs_CRIS3_YEDLYSNCK
122 и 79
0,011
0,675
CATD_VGFAEAAR_vs_CSH_ISLLLIESWLEPVR
57 и 81
0,011
0,676
PAPP1_DIPHWLNPTR_vs_CSH_AHQLAIDTYQEFEETYIPK
122 и 80
0,044
0,639
CATD_VGFAEAAR_vs_IBP3_FLNVLSPR
57 и 99
0,027
0,652
PAPP1_DIPHWLNPTR_vs_CSH_ISLLLIESWLEPVR
122 и 81
0,040
0,642
CATD_VGFAEAAR_vs_IBP3_YGQPLPGYTTK
57 и 100
0,036
0,644
PAPP1_DIPHWLNPTR_vs_LYAM1_SYYWIGIR
122 и 120
0,006
0,689
CATD_VGFAEAAR_vs_IGF2_GIVEECCFR
57 и 103
0,009
0,679
PAPP1_DIPHWLNPTR_vs_PGRP2_AGLLRPDYALLGHR
122 и 126
0,039
0,642
CATD_VGFAEAAR_vs_ITIH4_ILDDLSPR
57 и 112
0,034
0,646
PAPP1_DIPHWLNPTR_vs_PRG2_WNFAYWAAHQPWSR
122 и 129
0,008
0,681
CATD_VGFAEAAR_vs_LYAM1_SYYWIGIR
57 и 120
0,001
0,720
PAPP1_DIPHWLNPTR_vs_SHBG_IALGGLLFPASNLR
122 и 18
0,039
0,643
CATD_VGFAEAAR_vs_PGRP2_AGLLRPDYALLGHR
57 и 126
0,009
0,680
PAPP1_DIPHWLNPTR_vs_SOM2.CSH_NYGLLYCFR
122 и 138
0,039
0,642
CATD_VGFAEAAR_vs_SHBG_IALGGLLFPASNLR
57 и 18
0,029
0,651
PAPP1_DIPHWLNPTR_vs_SOM2.CSH_SVEGSCGF
122 и 139
0,005
0,693
CATD_VGFAEAAR_vs_SOM2.CSH_NYGLLYCFR
57 и 138
0,016
0,666
PEDF_LQSLFDSPDFSK_vs_CRIS3_YEDLYSNCK
124 и 79
0,047
0,637
CATD_VGFAEAAR_vs_SOM2.CSH_SVEGSCGF
57 и 139
0,011
0,675
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,005
0,693
CATD_VGFAEAAR_vs_SPRL1_VLTHSELAPLR
57 и 140
0,045
0,638
PEDF_TVQAVLTVPK_vs_CRIS3_AVSPPAR
125 и 78
0,042
0,640
CATD_VGFAEAAR_vs_TENX_LNWEAPPGAFDSFLLR
57 и 141
0,028
0,652
PEDF_TVQAVLTVPK_vs_CRIS3_YEDLYSNCK
125 и 79
0,027
0,653
CATD_VSTLPAITLK_vs_ALS_IRPHTFTGLSGLR
58 и 40
0,017
0,664
PEDF_TVQAVLTVPK_vs_LYAM1_SYYWIGIR
125 и 120
0,003
0,705
CATD_VSTLPAITLK_vs_C163A_INPASLDK
58 и 54
0,004
0,698
PEDF_TVQAVLTVPK_vs_PGRP2_AGLLRPDYALLGHR
125 и 126
0,030
0,649
CATD_VSTLPAITLK_vs_CRIS3_AVSPPAR
58 и 78
0,009
0,681
PSG2_IHPSYTNYR_vs_ALS_IRPHTFTGLSGLR
133 и 40
0,030
0,649
CATD_VSTLPAITLK_vs_CRIS3_YEDLYSNCK
58 и 79
0,004
0,698
PSG2_IHPSYTNYR_vs_CRIS3_AVSPPAR
133 и 78
0,004
0,700
CATD_VSTLPAITLK_vs_CSH_AHQLAIDTYQEFEETYIPK
58 и 80
0,011
0,676
PSG2_IHPSYTNYR_vs_CRIS3_YEDLYSNCK
133 и 79
0,004
0,700
CATD_VSTLPAITLK_vs_CSH_ISLLLIESWLEPVR
58 и 81
0,020
0,661
PSG2_IHPSYTNYR_vs_CSH_AHQLAIDTYQEFEETYIPK
133 и 80
0,032
0,648
CATD_VSTLPAITLK_vs_IBP1_VVESLAK
58 и 97
0,039
0,642
PSG2_IHPSYTNYR_vs_CSH_ISLLLIESWLEPVR
133 и 81
0,047
0,637
CATD_VSTLPAITLK_vs_IBP3_FLNVLSPR
58 и 99
0,012
0,673
PSG2_IHPSYTNYR_vs_FBLN1_TGYYFDGISR
133 и 86
0,040
0,641
CATD_VSTLPAITLK_vs_IBP3_YGQPLPGYTTK
58 и 100
0,019
0,661
PSG2_IHPSYTNYR_vs_IBP1_VVESLAK
133 и 97
0,040
0,641
CATD_VSTLPAITLK_vs_IGF2_GIVEECCFR
58 и 103
0,005
0,693
PSG2_IHPSYTNYR_vs_IBP2_LIQGAPTIR
133 и 98
0,009
0,681
CATD_VSTLPAITLK_vs_ITIH4_ILDDLSPR
58 и 112
0,033
0,647
PSG2_IHPSYTNYR_vs_IBP3_FLNVLSPR
133 и 99
0,028
0,652
CATD_VSTLPAITLK_vs_LYAM1_SYYWIGIR
58 и 120
0,001
0,727
PSG2_IHPSYTNYR_vs_IBP3_YGQPLPGYTTK
133 и 100
0,029
0,650
CATD_VSTLPAITLK_vs_PGRP2_AGLLRPDYALLGHR
58 и 126
0,004
0,700
PSG2_IHPSYTNYR_vs_IGF2_GIVEECCFR
133 и 103
0,017
0,664
CATD_VSTLPAITLK_vs_PSG3_VSAPSGTGHLPGLNPL
58 и 134
0,030
0,649
PSG2_IHPSYTNYR_vs_ITIH4_ILDDLSPR
133 и 112
0,016
0,666
CATD_VSTLPAITLK_vs_SHBG_IALGGLLFPASNLR
58 и 18
0,019
0,662
PSG2_IHPSYTNYR_vs_LYAM1_SYYWIGIR
133 и 120
0,001
0,729
CATD_VSTLPAITLK_vs_SOM2.CSH_NYGLLYCFR
58 и 138
0,025
0,654
PSG2_IHPSYTNYR_vs_PGRP2_AGLLRPDYALLGHR
133 и 126
0,007
0,686
CATD_VSTLPAITLK_vs_SOM2.CSH_SVEGSCGF
58 и 139
0,018
0,663
PSG2_IHPSYTNYR_vs_PSG3_VSAPSGTGHLPGLNPL
133 и 134
0,014
0,669
CATD_VSTLPAITLK_vs_TENX_LNWEAPPGAFDSFLLR
58 и 141
0,014
0,669
PSG2_IHPSYTNYR_vs_SHBG_IALGGLLFPASNLR
133 и 18
0,004
0,700
CBPN_EALIQFLEQVHQGIK_vs_LYAM1_SYYWIGIR
59 и 120
0,019
0,662
PSG2_IHPSYTNYR_vs_SOM2.CSH_SVEGSCGF
133 и 139
0,013
0,671
CBPN_NNANGVDLNR_vs_CRIS3_YEDLYSNCK
60 и 79
0,047
0,637
PSG2_IHPSYTNYR_vs_SPRL1_VLTHSELAPLR
133 и 140
0,028
0,652
CBPN_NNANGVDLNR_vs_LYAM1_SYYWIGIR
60 и 120
0,006
0,689
PSG2_IHPSYTNYR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
133 и 144
0,049
0,635
CD14_LTVGAAQVPAQLLVGALR_vs_CRIS3_AVSPPAR
61 и 78
0,046
0,637
PSG2_IHPSYTNYR_vs_VTDB_ELPEHTVK
133 и 147
0,021
0,659
CD14_LTVGAAQVPAQLLVGALR_vs_CRIS3_YEDLYSNCK
61 и 79
0,030
0,650
PTGDS_GPGEDFR_vs_CRIS3_AVSPPAR
137 и 78
0,042
0,640
CD14_LTVGAAQVPAQLLVGALR_vs_LYAM1_SYYWIGIR
61 и 120
0,003
0,708
PTGDS_GPGEDFR_vs_CRIS3_YEDLYSNCK
137 и 79
0,030
0,650
CD14_LTVGAAQVPAQLLVGALR_vs_PGRP2_AGLLRPDYALLGHR
61 и 126
0,009
0,679
PTGDS_GPGEDFR_vs_LYAM1_SYYWIGIR
137 и 120
0,011
0,676
CD14_LTVGAAQVPAQLLVGALR_vs_SHBG_IALGGLLFPASNLR
61 и 18
0,033
0,647
THBG_AVLHIGEK_vs_LYAM1_SYYWIGIR
143 и 120
0,024
0,655
CD14_LTVGAAQVPAQLLVGALR_vs_SOM2.CSH_SVEGSCGF
61 и 139
0,044
0,639
VTNC_GQYCYELDEK_vs_CRIS3_AVSPPAR
149 и 78
0,041
0,641
CD14_SWLAELQQWLKPGLK_vs_LYAM1_SYYWIGIR
62 и 120
0,009
0,679
VTNC_GQYCYELDEK_vs_CRIS3_YEDLYSNCK
149 и 79
0,023
0,657
CD14_SWLAELQQWLKPGLK_vs_PGRP2_AGLLRPDYALLGHR
62 и 126
0,022
0,657
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,001
0,719
CFAB_YGLVTYATYPK_vs_LYAM1_SYYWIGIR
64 и 120
0,038
0,643
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,021
0,659
CLUS_ASSIIDELFQDR_vs_CRIS3_AVSPPAR
67 и 78
0,029
0,651
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,047
0,637
CLUS_ASSIIDELFQDR_vs_CRIS3_YEDLYSNCK
67 и 79
0,018
0,663
VTNC_VDTVDPPYPR_vs_CRIS3_YEDLYSNCK
150 и 79
0,047
0,637
CLUS_ASSIIDELFQDR_vs_LYAM1_SYYWIGIR
67 и 120
0,003
0,703
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,007
0,685
CLUS_ASSIIDELFQDR_vs_PGRP2_AGLLRPDYALLGHR
67 и 126
0,035
0,645
Таблица 47: эффективность классификации по обращениям, недели 17, 18 и 19.
AUROC обращения для недель беременности от 17 0/7 до 19 6/7 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, без BMI-стратификации.
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн.
ROC_ AUC
A2GL_DLLLPQPDLR_vs_PSG3_VSAPSGTGHLPGLNPL
34 и 134
0,003
0,628
CO8B_QALEEFQK_vs_PSG3_VSAPSGTGHLPGLNPL
76 и 134
0,003
0,631
A2GL_DLLLPQPDLR_vs_SHBG_IALGGLLFPASNLR
34 и 18
0,021
0,600
CO8B_QALEEFQK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
76 и 144
0,003
0,628
AFAM_DADPDTFFAK_vs_PSG3_VSAPSGTGHLPGLNPL
37 и 134
0,007
0,617
F13B_GDTYPAELYITGSILR_vs_CHL1_VIAVNEVGR
84 и 66
0,020
0,601
AFAM_DADPDTFFAK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
37 и 144
0,016
0,606
F13B_GDTYPAELYITGSILR_vs_PSG3_VSAPSGTGHLPGLNPL
84 и 134
0,004
0,625
AFAM_HFQNLGK_vs_PSG3_VSAPSGTGHLPGLNPL
38 и 134
0,012
0,609
FETUA_FSVVYAK_vs_PSG3_VSAPSGTGHLPGLNPL
88 и 134
0,010
0,612
ANGT_DPTFIPAPIQAK_vs_CSH_AHQLAIDTYQEFEETYIPK
42 и 80
0,017
0,605
FETUA_HTLNQIDEVK_vs_PSG3_VSAPSGTGHLPGLNPL
89 и 134
0,016
0,605
ANGT_DPTFIPAPIQAK_vs_PSG3_VSAPSGTGHLPGLNPL
42 и 134
0,011
0,610
HABP2_FLNWIK_vs_CHL1_VIAVNEVGR
92 и 66
0,019
0,602
APOC3_GWVTDGFSSLK_vs_ALS_IRPHTFTGLSGLR
47 и 40
0,017
0,605
HABP2_FLNWIK_vs_FBLN1_TGYYFDGISR
92 и 86
0,021
0,601
APOC3_GWVTDGFSSLK_vs_C163A_INPASLDK
47 и 54
0,006
0,621
HABP2_FLNWIK_vs_IGF2_GIVEECCFR
92 и 103
0,012
0,609
APOC3_GWVTDGFSSLK_vs_CSH_AHQLAIDTYQEFEETYIPK
47 и 80
0,009
0,614
HABP2_FLNWIK_vs_PSG3_VSAPSGTGHLPGLNPL
92 и 134
0,001
0,640
APOC3_GWVTDGFSSLK_vs_IBP3_FLNVLSPR
47 и 99
0,013
0,608
HABP2_FLNWIK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
92 и 144
0,017
0,604
APOC3_GWVTDGFSSLK_vs_IBP3_YGQPLPGYTTK
47 и 100
0,017
0,604
HEMO_NFPSPVDAAFR_vs_PSG3_VSAPSGTGHLPGLNPL
93 и 134
0,005
0,622
APOC3_GWVTDGFSSLK_vs_IGF2_GIVEECCFR
47 и 103
0,013
0,608
IBP4_QCHPALDGQR_vs_CHL1_VIAVNEVGR
2 и 66
0,021
0,601
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,015
0,606
IBP4_QCHPALDGQR_vs_CSH_AHQLAIDTYQEFEETYIPK
2 и 80
0,017
0,604
APOC3_GWVTDGFSSLK_vs_NCAM1_GLGEISAASEFK
47 и 121
0,020
0,602
IBP4_QCHPALDGQR_vs_FBLN1_TGYYFDGISR
2 и 86
0,017
0,604
APOC3_GWVTDGFSSLK_vs_PSG3_VSAPSGTGHLPGLNPL
47 и 134
0,002
0,633
IBP4_QCHPALDGQR_vs_IGF2_GIVEECCFR
2 и 103
0,014
0,607
APOC3_GWVTDGFSSLK_vs_SHBG_IALGGLLFPASNLR
47 и 18
0,022
0,600
IBP4_QCHPALDGQR_vs_PSG3_VSAPSGTGHLPGLNPL
2 и 134
0,000
0,660
APOC3_GWVTDGFSSLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
47 и 144
0,009
0,614
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,011
0,612
APOH_ATVVYQGER_vs_CHL1_VIAVNEVGR
48 и 66
0,021
0,600
IBP6_GAQTLYVPNCDHR_vs_PSG3_VSAPSGTGHLPGLNPL
101 и 134
0,008
0,616
APOH_ATVVYQGER_vs_CSH_AHQLAIDTYQEFEETYIPK
48 и 80
0,009
0,614
IBP6_HLDSVLQQLQTEVYR_vs_PSG3_VSAPSGTGHLPGLNPL
102 и 134
0,006
0,621
APOH_ATVVYQGER_vs_FBLN1_TGYYFDGISR
48 и 86
0,011
0,610
INHBC_LDFHFSSDR_vs_IGF2_GIVEECCFR
107 и 103
0,015
0,606
APOH_ATVVYQGER_vs_IBP3_YGQPLPGYTTK
48 и 100
0,014
0,607
INHBC_LDFHFSSDR_vs_PSG3_VSAPSGTGHLPGLNPL
107 и 134
0,003
0,631
APOH_ATVVYQGER_vs_IGF2_GIVEECCFR
48 и 103
0,008
0,616
INHBC_LDFHFSSDR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
107 и 144
0,019
0,602
APOH_ATVVYQGER_vs_PSG3_VSAPSGTGHLPGLNPL
48 и 134
0,000
0,654
ITIH3_ALDLSLK_vs_PSG3_VSAPSGTGHLPGLNPL
111 и 134
0,018
0,603
APOH_ATVVYQGER_vs_SHBG_IALGGLLFPASNLR
48 и 18
0,010
0,612
ITIH3_ALDLSLK_vs_SHBG_IALGGLLFPASNLR
111 и 18
0,020
0,602
APOH_ATVVYQGER_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
48 и 144
0,010
0,613
KNG1_DIPTNSPELEETLTHTITK_vs_PSG3_VSAPSGTGHLPGLNPL
116 и 134
0,004
0,627
APOH_ATVVYQGER_vs_VTDB_ELPEHTVK
48 и 147
0,021
0,601
KNG1_QVVAGLNFR_vs_PSG3_VSAPSGTGHLPGLNPL
117 и 134
0,017
0,604
B2MG_VNHVTLSQPK_vs_PSG3_VSAPSGTGHLPGLNPL
51 и 134
0,010
0,613
LBP_ITGFLKPGK_vs_PSG3_VSAPSGTGHLPGLNPL
118 и 134
0,003
0,631
C1QB_VPGLYYFTYHASSR_vs_CSH_AHQLAIDTYQEFEETYIPK
55 и 80
0,009
0,613
LBP_ITLPDFTGDLR_vs_C163A_INPASLDK
119 и 54
0,012
0,609
C1QB_VPGLYYFTYHASSR_vs_PSG3_VSAPSGTGHLPGLNPL
55 и 134
0,010
0,613
LBP_ITLPDFTGDLR_vs_CSH_AHQLAIDTYQEFEETYIPK
119 и 80
0,019
0,602
C1QB_VPGLYYFTYHASSR_vs_SHBG_IALGGLLFPASNLR
55 и 18
0,013
0,608
LBP_ITLPDFTGDLR_vs_PSG3_VSAPSGTGHLPGLNPL
119 и 134
0,001
0,640
C1QB_VPGLYYFTYHASSR_vs_SOM2.CSH_NYGLLYCFR
55 и 138
0,021
0,600
LBP_ITLPDFTGDLR_vs_SHBG_IALGGLLFPASNLR
119 и 18
0,013
0,608
CD14_LTVGAAQVPAQLLVGALR_vs_CSH_AHQLAIDTYQEFEETYIPK
61 и 80
0,021
0,600
LBP_ITLPDFTGDLR_vs_SOM2.CSH_SVEGSCGF
119 и 139
0,021
0,602
CD14_LTVGAAQVPAQLLVGALR_vs_PSG3_VSAPSGTGHLPGLNPL
61 и 134
0,004
0,624
PEDF_LQSLFDSPDFSK_vs_PSG3_VSAPSGTGHLPGLNPL
124 и 134
0,002
0,633
CD14_SWLAELQQWLKPGLK_vs_CSH_AHQLAIDTYQEFEETYIPK
62 и 80
0,020
0,601
PEDF_TVQAVLTVPK_vs_CSH_AHQLAIDTYQEFEETYIPK
125 и 80
0,020
0,601
CD14_SWLAELQQWLKPGLK_vs_PSG3_VSAPSGTGHLPGLNPL
62 и 134
0,004
0,625
PEDF_TVQAVLTVPK_vs_PSG3_VSAPSGTGHLPGLNPL
125 и 134
0,002
0,636
CD14_SWLAELQQWLKPGLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
62 и 144
0,020
0,602
PEDF_TVQAVLTVPK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
125 и 144
0,018
0,603
CFAB_YGLVTYATYPK_vs_C163A_INPASLDK
64 и 54
0,007
0,617
PTGDS_GPGEDFR_vs_PSG3_VSAPSGTGHLPGLNPL
137 и 134
0,012
0,609
CFAB_YGLVTYATYPK_vs_CHL1_VIAVNEVGR
64 и 66
0,011
0,611
THBG_AVLHIGEK_vs_PSG3_VSAPSGTGHLPGLNPL
143 и 134
0,021
0,601
CFAB_YGLVTYATYPK_vs_CSH_AHQLAIDTYQEFEETYIPK
64 и 80
0,010
0,612
VTNC_GQYCYELDEK_vs_CHL1_VIAVNEVGR
149 и 66
0,013
0,608
CFAB_YGLVTYATYPK_vs_IGF2_GIVEECCFR
64 и 103
0,014
0,607
VTNC_GQYCYELDEK_vs_CSH_AHQLAIDTYQEFEETYIPK
149 и 80
0,012
0,609
CFAB_YGLVTYATYPK_vs_NCAM1_GLGEISAASEFK
64 и 121
0,009
0,614
VTNC_GQYCYELDEK_vs_FBLN1_TGYYFDGISR
149 и 86
0,010
0,612
CFAB_YGLVTYATYPK_vs_PSG3_VSAPSGTGHLPGLNPL
64 и 134
0,001
0,645
VTNC_GQYCYELDEK_vs_IBP3_YGQPLPGYTTK
149 и 100
0,016
0,605
CFAB_YGLVTYATYPK_vs_SHBG_IALGGLLFPASNLR
64 и 18
0,008
0,615
VTNC_GQYCYELDEK_vs_IGF2_GIVEECCFR
149 и 103
0,011
0,611
CFAB_YGLVTYATYPK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
64 и 144
0,001
0,639
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,009
0,613
CFAB_YGLVTYATYPK_vs_VTDB_ELPEHTVK
64 и 147
0,016
0,605
VTNC_GQYCYELDEK_vs_NCAM1_GLGEISAASEFK
149 и 121
0,010
0,612
CLUS_ASSIIDELFQDR_vs_PSG3_VSAPSGTGHLPGLNPL
67 и 134
0,006
0,619
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,008
0,616
CLUS_LFDSDPITVTVPVEVSR_vs_PSG3_VSAPSGTGHLPGLNPL
68 и 134
0,013
0,608
VTNC_GQYCYELDEK_vs_PSG3_VSAPSGTGHLPGLNPL
149 и 134
0,000
0,658
CO5_TLLPVSKPEIR_vs_PSG3_VSAPSGTGHLPGLNPL
70 и 134
0,001
0,641
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,004
0,625
CO5_TLLPVSKPEIR_vs_SHBG_IALGGLLFPASNLR
70 и 18
0,020
0,602
VTNC_GQYCYELDEK_vs_SOM2.CSH_SVEGSCGF
149 и 139
0,019
0,603
CO5_TLLPVSKPEIR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
70 и 144
0,011
0,610
VTNC_GQYCYELDEK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
149 и 144
0,002
0,633
CO5_VFQFLEK_vs_PSG3_VSAPSGTGHLPGLNPL
71 и 134
0,003
0,629
VTNC_GQYCYELDEK_vs_VTDB_ELPEHTVK
149 и 147
0,012
0,610
CO5_VFQFLEK_vs_SHBG_IALGGLLFPASNLR
71 и 18
0,018
0,604
VTNC_VDTVDPPYPR_vs_CHL1_VIAVNEVGR
150 и 66
0,018
0,603
CO5_VFQFLEK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
71 и 144
0,009
0,614
VTNC_VDTVDPPYPR_vs_CSH_AHQLAIDTYQEFEETYIPK
150 и 80
0,015
0,606
CO6_ALNHLPLEYNSALYSR_vs_CHL1_VIAVNEVGR
72 и 66
0,020
0,601
VTNC_VDTVDPPYPR_vs_IGF2_GIVEECCFR
150 и 103
0,018
0,604
CO6_ALNHLPLEYNSALYSR_vs_IGF2_GIVEECCFR
72 и 103
0,014
0,607
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,010
0,613
CO6_ALNHLPLEYNSALYSR_vs_PSG3_VSAPSGTGHLPGLNPL
72 и 134
0,002
0,634
VTNC_VDTVDPPYPR_vs_NCAM1_GLGEISAASEFK
150 и 121
0,011
0,610
CO6_ALNHLPLEYNSALYSR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
72 и 144
0,004
0,626
VTNC_VDTVDPPYPR_vs_PGRP2_AGLLRPDYALLGHR
150 и 126
0,021
0,601
CO8A_SLLQPNK_vs_CSH_AHQLAIDTYQEFEETYIPK
74 и 80
0,017
0,605
VTNC_VDTVDPPYPR_vs_PSG3_VSAPSGTGHLPGLNPL
150 и 134
0,000
0,656
CO8A_SLLQPNK_vs_PSG3_VSAPSGTGHLPGLNPL
74 и 134
0,002
0,632
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,010
0,612
CO8A_SLLQPNK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
74 и 144
0,004
0,625
VTNC_VDTVDPPYPR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
150 и 144
0,007
0,618
CO8B_QALEEFQK_vs_CSH_AHQLAIDTYQEFEETYIPK
76 и 80
0,012
0,609
VTNC_VDTVDPPYPR_vs_VTDB_ELPEHTVK
150 и 147
0,007
0,617
Таблица 48: эффективность классификации по обращениям, недели 17, 18 и 19.
AUROC обращения для недель беременности от 17 0/7 до 19 6/7 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, с BMI-стратификацией (>22≤37).
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
A2GL_DLLLPQPDLR_vs_PSG3_VSAPSGTGHLPGLNPL
34 и 134
0,030
0,616
F13B_GDTYPAELYITGSILR_vs_LYAM1_SYYWIGIR
84 и 120
0,040
0,609
A2GL_DLLLPQPDLR_vs_SHBG_IALGGLLFPASNLR
34 и 18
0,046
0,607
F13B_GDTYPAELYITGSILR_vs_PSG3_VSAPSGTGHLPGLNPL
84 и 134
0,027
0,618
AFAM_DADPDTFFAK_vs_PSG3_VSAPSGTGHLPGLNPL
37 и 134
0,030
0,616
HABP2_FLNWIK_vs_C163A_INPASLDK
92 и 54
0,028
0,617
AFAM_DADPDTFFAK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
37 и 144
0,020
0,624
HABP2_FLNWIK_vs_CHL1_VIAVN EVGR
92 и 66
0,017
0,628
ANGT_DPTFIPAPIQAK_vs_CSH_AHQLAIDTYQEFEETYIPK
42 и 80
0,024
0,621
HABP2_FLNWIK_vs_IGF2_GIVEEC CFR
92 и 103
0,023
0,622
ANGT_DPTFIPAPIQAK_vs_PSG3_VSAPSGTGHLPGLNPL
42 и 134
0,022
0,622
HABP2_FLNWIK_vs_LYAM1_SYYW IGIR
92 и 120
0,033
0,614
ANGT_DPTFIPAPIQAK_vs_SHBG_IALGGLLFPASNLR
42 и 18
0,040
0,610
HABP2_FLNWIK_vs_NCAM1_GLGEISAASEFK
92 и 121
0,036
0,612
ANGT_DPTFIPAPIQAK_vs_SOM2.CSH_SVEGSCGF
42 и 139
0,016
0,630
HABP2_FLNWIK_vs_PSG3_VSAPSGTGHLPGLNPL
92 и 134
0,008
0,642
ANGT_DPTFIPAPIQAK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
42 и 144
0,034
0,613
HABP2_FLNWIK_vs_SHBG_IALGGLLFPASNLR
92 и 18
0,044
0,608
APOC3_GWVTDGFSSLK_vs_C163A_INPASLDK
47 и 54
0,007
0,643
HABP2_FLNWIK_vs_SOM2.CSH_SVEGSCGF
92 и 139
0,034
0,615
APOC3_GWVTDGFSSLK_vs_CRIS3_AVSPPAR
47 и 78
0,044
0,607
HABP2_FLNWIK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
92 и 144
0,009
0,639
APOC3_GWVTDGFSSLK_vs_CRIS3_YEDLYSNCK
47 и 79
0,039
0,610
HABP2_FLNWIK_vs_VTDB_ELPEH TVK
92 и 147
0,021
0,623
APOC3_GWVTDGFSSLK_vs_IBP3_FLNVLSPR
47 и 99
0,045
0,607
IBP4_QCHPALDGQR_vs_CHL1_VIAVNEVGR
2 и 66
0,038
0,611
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,047
0,606
IBP4_QCHPALDGQR_vs_IGF2_GIVEECCFR
2 и 103
0,039
0,610
APOC3_GWVTDGFSSLK_vs_PSG3_VSAPSGTGHLPGLNPL
47 и 134
0,023
0,621
IBP4_QCHPALDGQR_vs_PSG3_VSAPSGTGHLPGLNPL
2 и 134
0,012
0,633
APOC3_GWVTDGFSSLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
47 и 144
0,050
0,605
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,044
0,607
APOH_ATVVYQGER_vs_CHL1_VIAVNEVGR
48 и 66
0,047
0,606
IBP4_QCHPALDGQR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
2 и 144
0,037
0,611
APOH_ATVVYQGER_vs_IBP3_YGQPLPGYTTK
48 и 100
0,043
0,608
INHBC_LDFHFSSDR_vs_C163A_INPASLDK
107 и 54
0,025
0,620
APOH_ATVVYQGER_vs_PSG3_VSAPSGTGHLPGLNPL
48 и 134
0,026
0,619
INHBC_LDFHFSSDR_vs_PSG3_VSAPSGTGHLPGLNPL
107 и 134
0,016
0,629
C1QB_VPGLYYFTYHASSR_vs_C163A_INPASLDK
55 и 54
0,048
0,605
INHBC_LDFHFSSDR_vs_TENX_LNWEAPPGAFDSFLLR
107 и 141
0,030
0,616
C1QB_VPGLYYFTYHASSR_vs_CHL1_VIAVNEVGR
55 и 66
0,033
0,614
INHBC_LDFHFSSDR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
107 и 144
0,049
0,605
C1QB_VPGLYYFTYHASSR_vs_CSH_AHQLAIDTYQEFEETYIPK
55 и 80
0,026
0,619
PEDF_LQSLFDSPDFSK_vs_C163A_INPASLDK
124 и 54
0,047
0,606
C1QB_VPGLYYFTYHASSR_vs_IBP3_YGQPLPGYTTK
55 и 100
0,049
0,605
PEDF_LQSLFDSPDFSK_vs_PSG3_VSAPSGTGHLPGLNPL
124 и 134
0,037
0,611
C1QB_VPGLYYFTYHASSR_vs_IGF2_GIVEECCFR
55 и 103
0,021
0,623
PEDF_TVQAVLTVPK_vs_C163A_INPASLDK
125 и 54
0,021
0,623
C1QB_VPGLYYFTYHASSR_vs_LYAM1_SYYWIGIR
55 и 120
0,024
0,621
PEDF_TVQAVLTVPK_vs_CHL1_VIAVNEVGR
125 и 66
0,026
0,619
C1QB_VPGLYYFTYHASSR_vs_PGRP2_AGLLRPDYALLGHR
55 и 126
0,020
0,624
PEDF_TVQAVLTVPK_vs_LYAM1_SYYWIGIR
125 и 120
0,038
0,611
C1QB_VPGLYYFTYHASSR_vs_PSG3_VSAPSGTGHLPGLNPL
55 и 134
0,022
0,622
PEDF_TVQAVLTVPK_vs_PSG3_VSAPSGTGHLPGLNPL
125 и 134
0,018
0,627
C1QB_VPGLYYFTYHASSR_vs_SHBG_IALGGLLFPASNLR
55 и 18
0,017
0,627
PEDF_TVQAVLTVPK_vs_TENX_LNWEAPPGAFDSFLLR
125 и 141
0,041
0,609
C1QB_VPGLYYFTYHASSR_vs_SOM2.CSH_NYGLLYCFR
55 и 138
0,040
0,610
PEDF_TVQAVLTVPK_vs_TENX_LSQLSVTDVTTSSLR
125 и 142
0,042
0,608
C1QB_VPGLYYFTYHASSR_vs_SOM2.CSH_SVEGSCGF
55 и 139
0,040
0,611
PEDF_TVQAVLTVPK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
125 и 144
0,028
0,617
C1QB_VPGLYYFTYHASSR_vs_TENX_LNWEAPPGAFDSFLLR
55 и 141
0,046
0,606
PRDX2_GLFIIDGK_vs_C163A_INPASLDK
128 и 54
0,039
0,610
C1QB_VPGLYYFTYHASSR_vs_TENX_LSQLSVTDVTTSSLR
55 и 142
0,038
0,611
PRDX2_GLFIIDGK_vs_PSG3_VSAPSGTGHLPGLNPL
128 и 134
0,047
0,606
C1QB_VPGLYYFTYHASSR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
55 и 144
0,033
0,614
PSG2_IHPSYTNYR_vs_C163A_INPASLDK
133 и 54
0,034
0,613
CBPN_EALIQFLEQVHQGIK_vs_LYAM1_SYYWIGIR
59 и 120
0,044
0,607
PSG2_IHPSYTNYR_vs_CHL1_VIAVNEVGR
133 и 66
0,048
0,606
CFAB_YGLVTYATYPK_vs_C163A_INPASLDK
64 и 54
0,008
0,642
PSG2_IHPSYTNYR_vs_CRIS3_AVSPPAR
133 и 78
0,045
0,607
CFAB_YGLVTYATYPK_vs_CHL1_VIAVNEVGR
64 и 66
0,030
0,616
PSG2_IHPSYTNYR_vs_CRIS3_YEDLYSNCK
133 и 79
0,049
0,605
CFAB_YGLVTYATYPK_vs_NCAM1_GLGEISAASEFK
64 и 121
0,022
0,622
PSG2_IHPSYTNYR_vs_FBLN1_TGYYFDGISR
133 и 86
0,031
0,615
CFAB_YGLVTYATYPK_vs_PSG3_VSAPSGTGHLPGLNPL
64 и 134
0,014
0,631
PSG2_IHPSYTNYR_vs_LYAM1_SYYWIGIR
133 и 120
0,024
0,620
CFAB_YGLVTYATYPK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
64 и 144
0,007
0,643
PSG2_IHPSYTNYR_vs_PSG1_FQLP GQK
133 и 131
0,047
0,606
CLUS_ASSIIDELFQDR_vs_PSG3_VSAPSGTGHLPGLNPL
67 и 134
0,048
0,605
PSG2_IHPSYTNYR_vs_PSG3_VSAPSGTGHLPGLNPL
133 и 134
0,028
0,617
CO5_TLLPVSKPEIR_vs_CHL1_VIAVNEVGR
70 и 66
0,019
0,625
PSG2_IHPSYTNYR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
133 и 135
0,026
0,619
CO5_TLLPVSKPEIR_vs_LYAM1_SYYWIGIR
70 и 120
0,014
0,632
PSG2_IHPSYTNYR_vs_SOM2.CSH_SVEGSCGF
133 и 139
0,043
0,609
CO5_TLLPVSKPEIR_vs_PGRP2_AGLLRPDYALLGHR
70 и 126
0,039
0,610
PSG2_IHPSYTNYR_vs_TENX_LSQLSVTDVTTSSLR
133 и 142
0,037
0,611
CO5_TLLPVSKPEIR_vs_PSG3_VSAPSGTGHLPGLNPL
70 и 134
0,006
0,647
VTNC_GQYCYELDEK_vs_C163A_INPASLDK
149 и 54
0,007
0,643
CO5_TLLPVSKPEIR_vs_SHBG_IALGGLLFPASNLR
70 и 18
0,037
0,611
VTNC_GQYCYELDEK_vs_CHL1_VIAVNEVGR
149 и 66
0,011
0,635
CO5_TLLPVSKPEIR_vs_TENX_LNWEAPPGAFDSFLLR
70 и 141
0,040
0,609
VTNC_GQYCYELDEK_vs_CRIS3_AVSPPAR
149 и 78
0,035
0,612
CO5_TLLPVSKPEIR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
70 и 144
0,012
0,635
VTNC_GQYCYELDEK_vs_CRIS3_YEDLYSNCK
149 и 79
0,042
0,609
CO5_TLLPVSKPEIR_vs_VTDB_ELPEHTVK
70 и 147
0,047
0,606
VTNC_GQYCYELDEK_vs_CSH_AHQLAIDTYQEFEETYIPK
149 и 80
0,040
0,609
CO5_VFQFLEK_vs_CHL1_VIAVNEVGR
71 и 66
0,036
0,612
VTNC_GQYCYELDEK_vs_IBP3_FLNVLSPR
149 и 99
0,046
0,606
CO5_VFQFLEK_vs_LYAM1_SYYWIGIR
71 и 120
0,021
0,623
VTNC_GQYCYELDEK_vs_IBP3_YGQPLPGYTTK
149 и 100
0,039
0,610
CO5_VFQFLEK_vs_PGRP2_AGLLRPDYALLGHR
71 и 126
0,035
0,612
VTNC_GQYCYELDEK_vs_IGF2_GIVEECCFR
149 и 103
0,035
0,612
CO5_VFQFLEK_vs_PSG3_VSAPSGTGHLPGLNPL
71 и 134
0,016
0,628
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,009
0,640
CO5_VFQFLEK_vs_SHBG_IALGGLLFPASNLR
71 и 18
0,034
0,613
VTNC_GQYCYELDEK_vs_NCAM1_GLGEISAASEFK
149 и 121
0,039
0,610
CO5_VFQFLEK_vs_TENX_LNWEAPPGAFDSFLLR
71 и 141
0,036
0,612
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,017
0,627
CO5_VFQFLEK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
71 и 144
0,012
0,634
VTNC_GQYCYELDEK_vs_PSG3_VSAPSGTGHLPGLNPL
149 и 134
0,004
0,652
CO6_ALNHLPLEYNSALYSR_vs_C163A_INPASLDK
72 и 54
0,043
0,608
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,021
0,623
CO6_ALNHLPLEYNSALYSR_vs_CHL1_VIAVNEVGR
72 и 66
0,014
0,631
VTNC_GQYCYELDEK_vs_SOM2.CSH_SVEGSCGF
149 и 139
0,028
0,618
CO6_ALNHLPLEYNSALYSR_vs_IGF2_GIVEECCFR
72 и 103
0,043
0,608
VTNC_GQYCYELDEK_vs_TENX_LNWEAPPGAFDSFLLR
149 и 141
0,042
0,609
CO6_ALNHLPLEYNSALYSR_vs_LYAM1_SYYWIGIR
72 и 120
0,026
0,619
VTNC_GQYCYELDEK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
149 и 144
0,003
0,658
CO6_ALNHLPLEYNSALYSR_vs_PSG3_VSAPSGTGHLPGLNPL
72 и 134
0,012
0,634
VTNC_GQYCYELDEK_vs_VTDB_ELPEHTVK
149 и 147
0,013
0,632
CO6_ALNHLPLEYNSALYSR_vs_TENX_LNWEAPPGAFDSFLLR
72 и 141
0,022
0,622
VTNC_VDTVDPPYPR_vs_C163A_INPASLDK
150 и 54
0,019
0,625
CO6_ALNHLPLEYNSALYSR_vs_TENX_LSQLSVTDVTTSSLR
72 и 142
0,036
0,612
VTNC_VDTVDPPYPR_vs_CHL1_VIAVNEVGR
150 и 66
0,022
0,622
CO6_ALNHLPLEYNSALYSR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
72 и 144
0,004
0,654
VTNC_VDTVDPPYPR_vs_CRIS3_AVSPPAR
150 и 78
0,047
0,606
CO8A_SLLQPNK_vs_CHL1_VIAVNEVGR
74 и 66
0,049
0,605
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,008
0,640
CO8A_SLLQPNK_vs_PSG3_VSAPSGTGHLPGLNPL
74 и 134
0,025
0,619
VTNC_VDTVDPPYPR_vs_NCAM1_GLGEISAASEFK
150 и 121
0,037
0,611
CO8A_SLLQPNK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
74 и 144
0,011
0,635
VTNC_VDTVDPPYPR_vs_PGRP2_AGLLRPDYALLGHR
150 и 126
0,044
0,607
CO8B_QALEEFQK_vs_PSG3_VSAPSGTGHLPGLNPL
76 и 134
0,032
0,614
VTNC_VDTVDPPYPR_vs_PSG3_VSAPSGTGHLPGLNPL
150 и 134
0,005
0,648
CO8B_QALEEFQK_vs_TENX_LNWEAPPGAFDSFLLR
76 и 141
0,041
0,609
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,036
0,612
CO8B_QALEEFQK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
76 и 144
0,012
0,634
VTNC_VDTVDPPYPR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
150 и 144
0,010
0,637
F13B_GDTYPAELYITGSILR_vs_CHL1_VIAVNEVGR
84 и 66
0,021
0,623
VTNC_VDTVDPPYPR_vs_VTDB_ELPEHTVK
150 и 147
0,013
0,633
F13B_GDTYPAELYITGSILR_vs_IGF2_GIVEECCFR
84 и 103
0,042
0,609
Таблица 49: эффективность классификации по обращениям, недели 17, 18 и 19.
AUROC обращения для недель беременности от 17 0/7 до 19 6/7 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, без BMI-стратификации.
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,036
0,648
FETUA_HTLNQIDEVK_vs_PSG9_LFIPQITR
89 и 136
0,024
0,659
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,046
0,641
HABP2_FLNWIK_vs_C163A_INPASLDK
92 и 54
0,032
0,651
ANGT_DPTFIPAPIQAK_vs_IGF2_GIVEECCFR
42 и 103
0,025
0,658
HABP2_FLNWIK_vs_IGF2_GIVEECCFR
92 и 103
0,014
0,674
ANGT_DPTFIPAPIQAK_vs_LYAM1_SYYWIGIR
42 и 120
0,038
0,646
HABP2_FLNWIK_vs_LYAM1_SYYWIGIR
92 и 120
0,020
0,664
APOH_ATVVYQGER_vs_LYAM1_SYYWIGIR
48 и 120
0,049
0,639
HABP2_FLNWIK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
92 и 144
0,021
0,663
B2MG_VNHVTLSQPK_vs_CRIS3_YEDLYSNCK
51 и 79
0,043
0,643
HEMO_NFPSPVDAAFR_vs_IGF2_GIVEECCFR
93 и 103
0,029
0,654
B2MG_VNHVTLSQPK_vs_IGF2_GIVEECCFR
51 и 103
0,039
0,646
IBP4_QCHPALDGQR_vs_CRIS3_AVSPPAR
2 и 78
0,034
0,649
B2MG_VNHVTLSQPK_vs_LYAM1_SYYWIGIR
51 и 120
0,034
0,650
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,017
0,668
BGH3_LTLLAPLNSVFK_vs_PSG9_LFIPQITR
52 и 136
0,049
0,639
IBP4_QCHPALDGQR_vs_IGF2_GIVEECCFR
2 и 103
0,015
0,672
CBPN_EALIQFLEQVHQGIK_vs_PSG9_LFIPQITR
59 и 136
0,047
0,640
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,012
0,676
CBPN_NNANGVDLNR_vs_LYAM1_SYYWIGIR
60 и 120
0,039
0,646
IBP6_GAQTLYVPNCDHR_vs_C163A_INPASLDK
101 и 54
0,031
0,652
CD14_LTVGAAQVPAQLLVGALR_vs_IGF2_GIVEECCFR
61 и 103
0,015
0,672
IBP6_GAQTLYVPNCDHR_vs_CRIS3_YEDLYSNCK
101 и 79
0,045
0,642
CD14_LTVGAAQVPAQLLVGALR_vs_LYAM1_SYYWIGIR
61 и 120
0,014
0,674
IBP6_GAQTLYVPNCDHR_vs_IGF2_GIVEECCFR
101 и 103
0,020
0,664
CD14_LTVGAAQVPAQLLVGALR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
61 и 144
0,021
0,663
IBP6_GAQTLYVPNCDHR_vs_LYAM1_SYYWIGIR
101 и 120
0,041
0,644
CD14_SWLAELQQWLKPGLK_vs_IGF2_GIVEECCFR
62 и 103
0,031
0,653
IBP6_HLDSVLQQLQTEVYR_vs_C163A_INPASLDK
102 и 54
0,027
0,656
CD14_SWLAELQQWLKPGLK_vs_LYAM1_SYYWIGIR
62 и 120
0,038
0,647
IBP6_HLDSVLQQLQTEVYR_vs_CRIS3_YEDLYSNCK
102 и 79
0,036
0,648
CD14_SWLAELQQWLKPGLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
62 и 144
0,036
0,648
IBP6_HLDSVLQQLQTEVYR_vs_IGF2_GIVEECCFR
102 и 103
0,026
0,658
CLUS_ASSIIDELFQDR_vs_ALS_IRPHTFTGLSGLR
67 и 40
0,043
0,643
IBP6_HLDSVLQQLQTEVYR_vs_LYAM1_SYYWIGIR
102 и 120
0,045
0,641
CLUS_ASSIIDELFQDR_vs_C163A_INPASLDK
67 и 54
0,041
0,644
KNG1_DIPTNSPELEETLTHTITK_vs_IGF2_GIVEECCFR
116 и 103
0,037
0,647
CLUS_ASSIIDELFQDR_vs_CRIS3_YEDLYSNCK
67 и 79
0,034
0,650
KNG1_DIPTNSPELEETLTHTITK_vs_LYAM1_SYYWIGIR
116 и 120
0,036
0,648
CLUS_ASSIIDELFQDR_vs_IBP3_FLNVLSPR
67 и 99
0,014
0,674
KNG1_QVVAGLNFR_vs_CRIS3_YEDLYSNCK
117 и 79
0,031
0,653
CLUS_ASSIIDELFQDR_vs_IBP3_YGQPLPGYTTK
67 и 100
0,010
0,681
KNG1_QVVAGLNFR_vs_IBP3_YGQPLPGYTTK
117 и 100
0,030
0,653
CLUS_ASSIIDELFQDR_vs_IGF2_GIVEECCFR
67 и 103
0,001
0,732
KNG1_QVVAGLNFR_vs_IGF2_GIVEECCFR
117 и 103
0,006
0,695
CLUS_ASSIIDELFQDR_vs_LYAM1_SYYWIGIR
67 и 120
0,004
0,702
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,013
0,676
CLUS_ASSIIDELFQDR_vs_VTDB_ELPEHTVK
67 и 147
0,034
0,650
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,047
0,640
CLUS_LFDSDPITVTVPVEVSR_vs_ALS_IRPHTFTGLSGLR
68 и 40
0,039
0,645
PEDF_TVQAVLTVPK_vs_CRIS3_YEDLYSNCK
125 и 79
0,037
0,647
CLUS_LFDSDPITVTVPVEVSR_vs_C163A_INPASLDK
68 и 54
0,037
0,647
PEDF_TVQAVLTVPK_vs_IGF2_GIVEECCFR
125 и 103
0,042
0,643
CLUS_LFDSDPITVTVPVEVSR_vs_CRIS3_AVSPPAR
68 и 78
0,027
0,656
PEDF_TVQAVLTVPK_vs_LYAM1_SYYWIGIR
125 и 120
0,019
0,665
CLUS_LFDSDPITVTVPVEVSR_vs_CRIS3_YEDLYSNCK
68 и 79
0,012
0,678
PSG2_IHPSYTNYR_vs_ALS_IRPHTFTGLSGLR
133 и 40
0,029
0,654
CLUS_LFDSDPITVTVPVEVSR_vs_CSH_AHQLAIDTYQEFEETYIPK
68 и 80
0,050
0,638
PSG2_IHPSYTNYR_vs_C163A_INPASLDK
133 и 54
0,016
0,670
CLUS_LFDSDPITVTVPVEVSR_vs_IBP3_FLNVLSPR
68 и 99
0,005
0,700
PSG2_IHPSYTNYR_vs_CHL1_VIAVNEVGR
133 и 66
0,049
0,639
CLUS_LFDSDPITVTVPVEVSR_vs_IBP3_YGQPLPGYTTK
68 и 100
0,003
0,707
PSG2_IHPSYTNYR_vs_CRIS3_AVSPPAR
133 и 78
0,008
0,687
CLUS_LFDSDPITVTVPVEVSR_vs_IGF2_GIVEECCFR
68 и 103
0,000
0,749
PSG2_IHPSYTNYR_vs_CRIS3_YEDLYSNCK
133 и 79
0,005
0,697
CLUS_LFDSDPITVTVPVEVSR_vs_ITIH4_ILDDLSPR
68 и 112
0,030
0,653
PSG2_IHPSYTNYR_vs_CSH_AHQLAIDTYQEFEETYIPK
133 и 80
0,011
0,680
CLUS_LFDSDPITVTVPVEVSR_vs_LYAM1_SYYWIGIR
68 и 120
0,004
0,705
PSG2_IHPSYTNYR_vs_CSH_ISLLLIESWLEPVR
133 и 81
0,026
0,658
CLUS_LFDSDPITVTVPVEVSR_vs_TENX_LNWEAPPGAFDSFLLR
68 и 141
0,028
0,655
PSG2_IHPSYTNYR_vs_FBLN1_TGYYFDGISR
133 и 86
0,007
0,689
CLUS_LFDSDPITVTVPVEVSR_vs_TENX_LSQLSVTDVTTSSLR
68 и 142
0,045
0,641
PSG2_IHPSYTNYR_vs_IBP1_VVESLAK
133 и 97
0,027
0,656
CLUS_LFDSDPITVTVPVEVSR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
68 и 144
0,035
0,648
PSG2_IHPSYTNYR_vs_IBP2_LIQGAPTIR
133 и 98
0,031
0,652
CLUS_LFDSDPITVTVPVEVSR_vs_VTDB_ELPEHTVK
68 и 147
0,015
0,672
PSG2_IHPSYTNYR_vs_IBP3_FLNVLSPR
133 и 99
0,021
0,663
CO5_TLLPVSKPEIR_vs_CRIS3_YEDLYSNCK
70 и 79
0,047
0,640
PSG2_IHPSYTNYR_vs_IBP3_YGQPLPGYTTK
133 и 100
0,016
0,669
CO5_TLLPVSKPEIR_vs_IGF2_GIVEECCFR
70 и 103
0,036
0,648
PSG2_IHPSYTNYR_vs_IGF2_GIVEECCFR
133 и 103
0,010
0,681
CO5_TLLPVSKPEIR_vs_LYAM1_SYYWIGIR
70 и 120
0,021
0,663
PSG2_IHPSYTNYR_vs_ITIH4_ILDDLSPR
133 и 112
0,027
0,656
CO5_VFQFLEK_vs_LYAM1_SYYWIGIR
71 и 120
0,039
0,645
PSG2_IHPSYTNYR_vs_LYAM1_SYYWIGIR
133 и 120
0,004
0,702
CO6_ALNHLPLEYNSALYSR_vs_IGF2_GIVEECCFR
72 и 103
0,012
0,677
PSG2_IHPSYTNYR_vs_NCAM1_GLGEISAASEFK
133 и 121
0,021
0,663
CO6_ALNHLPLEYNSALYSR_vs_LYAM1_SYYWIGIR
72 и 120
0,007
0,690
PSG2_IHPSYTNYR_vs_PSG3_VSAPSGTGHLPGLNPL
133 и 134
0,013
0,676
CO6_ALNHLPLEYNSALYSR_vs_TENX_LNWEAPPGAFDSFLLR
72 и 141
0,038
0,647
PSG2_IHPSYTNYR_vs_SHBG_IALGGLLFPASNLR
133 и 18
0,025
0,658
CO6_ALNHLPLEYNSALYSR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
72 и 144
0,017
0,669
PSG2_IHPSYTNYR_vs_SOM2.CSH_NYGLLYCFR
133 и 138
0,019
0,665
CO8A_SLLQPNK_vs_IGF2_GIVEECCFR
74 и 103
0,036
0,648
PSG2_IHPSYTNYR_vs_SOM2.CSH_SVEGSCGF
133 и 139
0,034
0,650
CO8A_SLLQPNK_vs_LYAM1_SYYWIGIR
74 и 120
0,021
0,663
PSG2_IHPSYTNYR_vs_SPRL1_VLTHSELAPLR
133 и 140
0,024
0,660
CO8A_SLLQPNK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
74 и 144
0,046
0,641
PSG2_IHPSYTNYR_vs_TENX_LNWEAPPGAFDSFLLR
133 и 141
0,017
0,668
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
82 и 135
0,027
0,656
PSG2_IHPSYTNYR_vs_TENX_LSQLSVTDVTTSSLR
133 и 142
0,014
0,674
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PSG9_LFIPQITR
82 и 136
0,016
0,670
PSG2_IHPSYTNYR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
133 и 144
0,019
0,666
ENPP2_TYLHTYESEI_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
83 и 135
0,034
0,650
PSG2_IHPSYTNYR_vs_VTDB_ELPEHTVK
133 и 147
0,036
0,648
ENPP2_TYLHTYESEI_vs_PSG9_LFIPQITR
83 и 136
0,023
0,661
PTGDS_GPGEDFR_vs_CRIS3_YEDLYSNCK
137 и 79
0,043
0,643
F13B_GDTYPAELYITGSILR_vs_CRIS3_YEDLYSNCK
84 и 79
0,032
0,651
PTGDS_GPGEDFR_vs_IGF2_GIVEECCFR
137 и 103
0,046
0,641
F13B_GDTYPAELYITGSILR_vs_IBP3_YGQPLPGYTTK
84 и 100
0,049
0,639
VTNC_GQYCYELDEK_vs_IGF2_GIVEECCFR
149 и 103
0,025
0,658
F13B_GDTYPAELYITGSILR_vs_IGF2_GIVEECCFR
84 и 103
0,008
0,686
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,014
0,674
F13B_GDTYPAELYITGSILR_vs_LYAM1_SYYWIGIR
84 и 120
0,009
0,686
VTNC_VDTVDPPYPR_vs_IGF2_GIVEECCFR
150 и 103
0,043
0,643
FETUA_FSVVYAK_vs_PSG9_LFIPQITR
88 и 136
0,034
0,650
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,020
0,664
FETUA_HTLNQIDEVK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
89 и 135
0,037
0,647
Таблица 50: эффективность классификации по обращениям, недели 17, 18 и 19.
AUROC обращения для недель беременности от 17 0/7 до 19 6/7 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, с BMI-стратификацией (>22≤37).
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,012
0,718
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,013
0,715
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,023
0,696
IBP6_HLDSVLQQLQTEVYR_vs_IGF2_GIVEECCFR
102 и 103
0,049
0,670
ANGT_DPTFIPAPIQAK_vs_IGF2_GIVEECCFR
42 и 103
0,019
0,702
KNG1_DIPTNSPELEETLTHTITK_vs_IGF2_GIVEECCFR
116 и 103
0,019
0,704
ANGT_DPTFIPAPIQAK_vs_LYAM1_SYYWIGIR
42 и 120
0,035
0,682
KNG1_DIPTNSPELEETLTHTITK_vs_LYAM1_SYYWIGIR
116 и 120
0,030
0,688
APOH_ATVVYQGER_vs_IGF2_GIVEECCFR
48 и 103
0,040
0,678
KNG1_QVVAGLNFR_vs_CRIS3_YEDLYSNCK
117 и 79
0,037
0,681
B2MG_VNHVTLSQPK_vs_IGF2_GIVEECCFR
51 и 103
0,033
0,684
KNG1_QVVAGLNFR_vs_IBP3_FLNVLSPR
117 и 99
0,019
0,703
B2MG_VNHVTLSQPK_vs_LYAM1_SYYWIGIR
51 и 120
0,037
0,680
KNG1_QVVAGLNFR_vs_IBP3_YGQPLPGYTTK
117 и 100
0,024
0,696
CBPN_NNANGVDLNR_vs_LYAM1_SYYWIGIR
60 и 120
0,048
0,671
KNG1_QVVAGLNFR_vs_IGF2_GIVEECCFR
117 и 103
0,002
0,763
CD14_LTVGAAQVPAQLLVGALR_vs_IGF2_GIVEECCFR
61 и 103
0,020
0,702
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,014
0,713
CD14_LTVGAAQVPAQLLVGALR_vs_LYAM1_SYYWIGIR
61 и 120
0,020
0,702
PAPP1_DIPHWLNPTR_vs_C163A_INPASLDK
122 и 54
0,049
0,670
CLUS_ASSIIDELFQDR_vs_CRIS3_YEDLYSNCK
67 и 79
0,038
0,679
PAPP1_DIPHWLNPTR_vs_CRIS3_AVSPPAR
122 и 78
0,034
0,683
CLUS_ASSIIDELFQDR_vs_IBP3_FLNVLSPR
67 и 99
0,034
0,684
PAPP1_DIPHWLNPTR_vs_CRIS3_YEDLYSNCK
122 и 79
0,025
0,693
CLUS_ASSIIDELFQDR_vs_IGF2_GIVEECCFR
67 и 103
0,004
0,751
PAPP1_DIPHWLNPTR_vs_LYAM1_SYYWIGIR
122 и 120
0,036
0,681
CLUS_ASSIIDELFQDR_vs_LYAM1_SYYWIGIR
67 и 120
0,010
0,722
PEDF_TVQAVLTVPK_vs_IGF2_GIVEECCFR
125 и 103
0,031
0,687
CLUS_LFDSDPITVTVPVEVSR_vs_CRIS3_YEDLYSNCK
68 и 79
0,031
0,687
PEDF_TVQAVLTVPK_vs_LYAM1_SYYWIGIR
125 и 120
0,022
0,699
CLUS_LFDSDPITVTVPVEVSR_vs_IBP3_FLNVLSPR
68 и 99
0,033
0,685
PSG2_IHPSYTNYR_vs_ALS_IRPHTFTGLSGLR
133 и 40
0,018
0,705
CLUS_LFDSDPITVTVPVEVSR_vs_IGF2_GIVEECCFR
68 и 103
0,008
0,731
PSG2_IHPSYTNYR_vs_C163A_INPASLDK
133 и 54
0,010
0,723
CLUS_LFDSDPITVTVPVEVSR_vs_LYAM1_SYYWIGIR
68 и 120
0,028
0,690
PSG2_IHPSYTNYR_vs_CHL1_VIAVNEVGR
133 и 66
0,042
0,676
CO5_TLLPVSKPEIR_vs_IGF2_GIVEECCFR
70 и 103
0,029
0,689
PSG2_IHPSYTNYR_vs_CRIS3_AVSPPAR
133 и 78
0,003
0,758
CO5_TLLPVSKPEIR_vs_LYAM1_SYYWIGIR
70 и 120
0,015
0,710
PSG2_IHPSYTNYR_vs_CRIS3_YEDLYSNCK
133 и 79
0,003
0,759
CO5_VFQFLEK_vs_LYAM1_SYYWIGIR
71 и 120
0,045
0,673
PSG2_IHPSYTNYR_vs_CSH_AHQLAIDTYQEFEETYIPK
133 и 80
0,014
0,713
CO6_ALNHLPLEYNSALYSR_vs_IGF2_GIVEECCFR
72 и 103
0,008
0,731
PSG2_IHPSYTNYR_vs_CSH_ISLLLIESWLEPVR
133 и 81
0,029
0,690
CO6_ALNHLPLEYNSALYSR_vs_LYAM1_SYYWIGIR
72 и 120
0,011
0,720
PSG2_IHPSYTNYR_vs_FBLN1_TGYYFDGISR
133 и 86
0,014
0,712
CO8A_SLLQPNK_vs_IGF2_GIVEECCFR
74 и 103
0,049
0,671
PSG2_IHPSYTNYR_vs_IBP2_LIQGAPTIR
133 и 98
0,038
0,679
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_ALS_IRPHTFTGLSGLR
82 и 40
0,033
0,684
PSG2_IHPSYTNYR_vs_IBP3_FLNVLSPR
133 и 99
0,007
0,732
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_FBLN1_TGYYFDGISR
82 и 86
0,020
0,701
PSG2_IHPSYTNYR_vs_IBP3_YGQPLPGYTTK
133 и 100
0,010
0,722
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_ITIH4_ILDDLSPR
82 и 112
0,036
0,681
PSG2_IHPSYTNYR_vs_IGF2_GIVEECCFR
133 и 103
0,003
0,754
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_NCAM1_GLGEISAASEFK
82 и 121
0,026
0,693
PSG2_IHPSYTNYR_vs_ITIH4_ILDDLSPR
133 и 112
0,015
0,710
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PRG2_WNFAYWAAHQPWSR
82 и 129
0,042
0,676
PSG2_IHPSYTNYR_vs_LYAM1_SYYWIGIR
133 и 120
0,002
0,773
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
82 и 135
0,019
0,702
PSG2_IHPSYTNYR_vs_NCAM1_GLGEISAASEFK
133 и 121
0,035
0,683
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PSG9_LFIPQITR
82 и 136
0,006
0,739
PSG2_IHPSYTNYR_vs_PGRP2_AGLLRPDYALLGHR
133 и 126
0,021
0,700
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_VTDB_ELPEHTVK
82 и 147
0,016
0,709
PSG2_IHPSYTNYR_vs_PSG3_VSAPSGTGHLPGLNPL
133 и 134
0,007
0,733
ENPP2_TYLHTYESEI_vs_ALS_IRPHTFTGLSGLR
83 и 40
0,042
0,676
PSG2_IHPSYTNYR_vs_SHBG_IALGGLLFPASNLR
133 и 18
0,028
0,691
ENPP2_TYLHTYESEI_vs_FBLN1_TGYYFDGISR
83 и 86
0,025
0,693
PSG2_IHPSYTNYR_vs_SOM2.CSH_NYGLLYCFR
133 и 138
0,021
0,699
ENPP2_TYLHTYESEI_vs_ITIH4_ILDDLSPR
83 и 112
0,043
0,675
PSG2_IHPSYTNYR_vs_SOM2.CSH_SVEGSCGF
133 и 139
0,023
0,697
ENPP2_TYLHTYESEI_vs_NCAM1_GLGEISAASEFK
83 и 121
0,030
0,688
PSG2_IHPSYTNYR_vs_SPRL1_VLTHSELAPLR
133 и 140
0,042
0,676
ENPP2_TYLHTYESEI_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
83 и 135
0,027
0,692
PSG2_IHPSYTNYR_vs_TENX_LNWEAPPGAFDSFLLR
133 и 141
0,016
0,708
ENPP2_TYLHTYESEI_vs_PSG9_LFIPQITR
83 и 136
0,009
0,727
PSG2_IHPSYTNYR_vs_TENX_LSQLSVTDVTTSSLR
133 и 142
0,011
0,720
ENPP2_TYLHTYESEI_vs_VTDB_ELPEHTVK
83 и 147
0,015
0,712
PSG2_IHPSYTNYR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
133 и 144
0,031
0,687
F13B_GDTYPAELYITGSILR_vs_CRIS3_AVSPPAR
84 и 78
0,043
0,675
PSG2_IHPSYTNYR_vs_VTDB_ELPEHTVK
133 и 147
0,017
0,707
F13B_GDTYPAELYITGSILR_vs_CRIS3_YEDLYSNCK
84 и 79
0,030
0,688
PTGDS_GPGEDFR_vs_CRIS3_YEDLYSNCK
137 и 79
0,042
0,676
F13B_GDTYPAELYITGSILR_vs_IBP3_FLNVLSPR
84 и 99
0,040
0,678
PTGDS_GPGEDFR_vs_IGF2_GIVEECCFR
137 и 103
0,021
0,700
F13B_GDTYPAELYITGSILR_vs_IGF2_GIVEECCFR
84 и 103
0,004
0,750
VTNC_GQYCYELDEK_vs_IGF2_GIVEECCFR
149 и 103
0,018
0,704
F13B_GDTYPAELYITGSILR_vs_LYAM1_SYYWIGIR
84 и 120
0,012
0,718
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,017
0,707
HEMO_NFPSPVDAAFR_vs_IGF2_GIVEECCFR
93 и 103
0,045
0,673
VTNC_VDTVDPPYPR_vs_IGF2_GIVEECCFR
150 и 103
0,048
0,671
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,035
0,682
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,040
0,677
IBP4_QCHPALDGQR_vs_IGF2_GIVEECCFR
2 и 103
0,006
0,737
Таблица 51: эффективность классификации по обращениям, недели 18, 19 и 20.
AUROC обращения для недель беременности от 18 0/7 до 20 6/7 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, без BMI-стратификации.
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
A2GL_DLLLPQPDLR_vs_PSG3_VSAPSGTGHLPGLNPL
34 и 134
0,001
0,633
HABP2_FLNWIK_vs_CHL1_VIAVNE VGR
92 и 66
0,006
0,613
A2GL_DLLLPQPDLR_vs_SHBG_IALGGLLFPASNLR
34 и 18
0,008
0,608
HABP2_FLNWIK_vs_FBLN1_TGYYFDGISR
92 и 86
0,005
0,615
AFAM_DADPDTFFAK_vs_PSG3_VSAPSGTGHLPGLNPL
37 и 134
0,001
0,630
HABP2_FLNWIK_vs_IGF2_GIVEECCFR
92 и 103
0,005
0,613
AFAM_HFQNLGK_vs_PRG2_WNFAYWAAHQPWSR
38 и 129
0,010
0,605
HABP2_FLNWIK_vs_LYAM1_SYYWIGIR
92 и 120
0,014
0,601
AFAM_HFQNLGK_vs_PSG3_VSAPSGTGHLPGLNPL
38 и 134
0,001
0,636
HABP2_FLNWIK_vs_PRG2_WNFAYWAAHQPWSR
92 и 129
0,005
0,615
AFAM_HFQNLGK_vs_SHBG_IALGGLLFPASNLR
38 и 18
0,013
0,602
HABP2_FLNWIK_vs_PSG3_VSAPSGTGHLPGLNPL
92 и 134
0,000
0,658
AFAM_HFQNLGK_vs_TENX_LNWEAPPGAFDSFLLR
38 и 141
0,007
0,610
HABP2_FLNWIK_vs_SHBG_IALGGLLFPASNLR
92 и 18
0,004
0,618
ANGT_DPTFIPAPIQAK_vs_FBLN1_TGYYFDGISR
42 и 86
0,008
0,608
HABP2_FLNWIK_vs_TENX_LNWEAPPGAFDSFLLR
92 и 141
0,001
0,631
ANGT_DPTFIPAPIQAK_vs_PRG2_WNFAYWAAHQPWSR
42 и 129
0,010
0,605
HABP2_FLNWIK_vs_TENX_LSQLSVTDVTTSSLR
92 и 142
0,003
0,621
ANGT_DPTFIPAPIQAK_vs_PSG3_VSAPSGTGHLPGLNPL
42 и 134
0,001
0,635
HABP2_FLNWIK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
92 и 144
0,005
0,613
ANGT_DPTFIPAPIQAK_vs_SHBG_IALGGLLFPASNLR
42 и 18
0,005
0,614
HABP2_FLNWIK_vs_VTDB_ELPEH TVK
92 и 147
0,008
0,609
APOC3_GWVTDGFSSLK_vs_ALS_IRPHTFTGLSGLR
47 и 40
0,004
0,618
HEMO_NFPSPVDAAFR_vs_PSG3_VSAPSGTGHLPGLNPL
93 и 134
0,002
0,625
APOC3_GWVTDGFSSLK_vs_C163A_INPASLDK
47 и 54
0,001
0,638
HEMO_NFPSPVDAAFR_vs_SHBG_IALGGLLFPASNLR
93 и 18
0,007
0,610
APOC3_GWVTDGFSSLK_vs_CHL1_VIAVNEVGR
47 и 66
0,003
0,620
HEMO_NFPSPVDAAFR_vs_TENX_LNWEAPPGAFDSFLLR
93 и 141
0,006
0,612
APOC3_GWVTDGFSSLK_vs_CRIS3_AVSPPAR
47 и 78
0,001
0,631
IBP4_QCHPALDGQR_vs_CHL1_VIAVNEVGR
2 и 66
0,003
0,619
APOC3_GWVTDGFSSLK_vs_CRIS3_YEDLYSNCK
47 и 79
0,001
0,635
IBP4_QCHPALDGQR_vs_CRIS3_AVSPPAR
2 и 78
0,009
0,607
APOC3_GWVTDGFSSLK_vs_CSH_AHQLAIDTYQEFEETYIPK
47 и 80
0,002
0,627
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,004
0,618
APOC3_GWVTDGFSSLK_vs_CSH_ISLLLIESWLEPVR
47 и 81
0,006
0,613
IBP4_QCHPALDGQR_vs_FBLN1_TGYYFDGISR
2 и 86
0,002
0,629
APOC3_GWVTDGFSSLK_vs_FBLN1_TGYYFDGISR
47 и 86
0,001
0,634
IBP4_QCHPALDGQR_vs_IGF2_GIVEECCFR
2 и 103
0,012
0,602
APOC3_GWVTDGFSSLK_vs_IBP2_LIQGAPTIR
47 и 98
0,011
0,603
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,008
0,608
APOC3_GWVTDGFSSLK_vs_IBP3_FLNVLSPR
47 и 99
0,001
0,630
IBP4_QCHPALDGQR_vs_PGRP2_AGLLRPDYALLGHR
2 и 126
0,009
0,606
APOC3_GWVTDGFSSLK_vs_IBP3_YGQPLPGYTTK
47 и 100
0,002
0,623
IBP4_QCHPALDGQR_vs_PRG2_WNFAYWAAHQPWSR
2 и 129
0,004
0,619
APOC3_GWVTDGFSSLK_vs_IGF2_GIVEECCFR
47 и 103
0,001
0,633
IBP4_QCHPALDGQR_vs_PSG3_VSAPSGTGHLPGLNPL
2 и 134
0,000
0,688
APOC3_GWVTDGFSSLK_vs_ITIH4_ILDDLSPR
47 и 112
0,003
0,623
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,001
0,639
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,001
0,637
IBP4_QCHPALDGQR_vs_SPRL1_VLTHSELAPLR
2 и 140
0,010
0,605
APOC3_GWVTDGFSSLK_vs_NCAM1_GLGEISAASEFK
47 и 121
0,004
0,617
IBP4_QCHPALDGQR_vs_TENX_LNWEAPPGAFDSFLLR
2 и 141
0,001
0,634
APOC3_GWVTDGFSSLK_vs_PGRP2_AGLLRPDYALLGHR
47 и 126
0,003
0,620
IBP4_QCHPALDGQR_vs_TENX_LSQLSVTDVTTSSLR
2 и 142
0,006
0,612
APOC3_GWVTDGFSSLK_vs_PRG2_WNFAYWAAHQPWSR
47 и 129
0,001
0,637
IBP4_QCHPALDGQR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
2 и 144
0,003
0,619
APOC3_GWVTDGFSSLK_vs_PSG1_FQLPGQK
47 и 131
0,009
0,606
IBP4_QCHPALDGQR_vs_VTDB_ELPEHTVK
2 и 147
0,007
0,610
APOC3_GWVTDGFSSLK_vs_PSG3_VSAPSGTGHLPGLNPL
47 и 134
0,000
0,662
IBP6_GAQTLYVPNCDHR_vs_PSG3_VSAPSGTGHLPGLNPL
101 и 134
0,008
0,608
APOC3_GWVTDGFSSLK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
47 и 135
0,005
0,615
IBP6_HLDSVLQQLQTEVYR_vs_PSG3_VSAPSGTGHLPGLNPL
102 и 134
0,006
0,612
APOC3_GWVTDGFSSLK_vs_PSG9_LFIPQITR
47 и 136
0,006
0,612
INHBC_LDFHFSSDR_vs_CHL1_VIAVNEVGR
107 и 66
0,005
0,614
APOC3_GWVTDGFSSLK_vs_SHBG_IALGGLLFPASNLR
47 и 18
0,001
0,632
INHBC_LDFHFSSDR_vs_FBLN1_TGYYFDGISR
107 и 86
0,004
0,616
APOC3_GWVTDGFSSLK_vs_SOM2.CSH_NYGLLYCFR
47 и 138
0,006
0,611
INHBC_LDFHFSSDR_vs_IGF2_GIVEECCFR
107 и 103
0,003
0,622
APOC3_GWVTDGFSSLK_vs_SOM2.CSH_SVEGSCGF
47 и 139
0,008
0,609
INHBC_LDFHFSSDR_vs_PGRP2_AGLLRPDYALLGHR
107 и 126
0,008
0,609
APOC3_GWVTDGFSSLK_vs_SPRL1_VLTHSELAPLR
47 и 140
0,004
0,618
INHBC_LDFHFSSDR_vs_PRG2_WNFAYWAAHQPWSR
107 и 129
0,002
0,624
APOC3_GWVTDGFSSLK_vs_TENX_LNWEAPPGAFDSFLLR
47 и 141
0,001
0,634
INHBC_LDFHFSSDR_vs_PSG3_VSAPSGTGHLPGLNPL
107 и 134
0,000
0,659
APOC3_GWVTDGFSSLK_vs_TENX_LSQLSVTDVTTSSLR
47 и 142
0,001
0,630
INHBC_LDFHFSSDR_vs_SHBG_IALGGLLFPASNLR
107 и 18
0,003
0,620
APOC3_GWVTDGFSSLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
47 и 144
0,001
0,638
INHBC_LDFHFSSDR_vs_SPRL1_VLTHSELAPLR
107 и 140
0,008
0,608
APOC3_GWVTDGFSSLK_vs_VTDB_ELPEHTVK
47 и 147
0,001
0,630
INHBC_LDFHFSSDR_vs_TENX_LNWEAPPGAFDSFLLR
107 и 141
0,001
0,637
APOH_ATVVYQGER_vs_FBLN1_TGYYFDGISR
48 и 86
0,005
0,614
INHBC_LDFHFSSDR_vs_TENX_LSQLSVTDVTTSSLR
107 и 142
0,002
0,624
APOH_ATVVYQGER_vs_PRG2_WNFAYWAAHQPWSR
48 и 129
0,013
0,601
INHBC_LDFHFSSDR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
107 и 144
0,003
0,620
APOH_ATVVYQGER_vs_PSG3_VSAPSGTGHLPGLNPL
48 и 134
0,000
0,649
INHBC_LDFHFSSDR_vs_VTDB_ELPEHTVK
107 и 147
0,008
0,608
APOH_ATVVYQGER_vs_SHBG_IALGGLLFPASNLR
48 и 18
0,006
0,613
ITIH3_ALDLSLK_vs_PSG3_VSAPSGTGHLPGLNPL
111 и 134
0,006
0,612
APOH_ATVVYQGER_vs_TENX_LNWEAPPGAFDSFLLR
48 и 141
0,008
0,607
ITIH3_ALDLSLK_vs_SHBG_IALGGLLFPASNLR
111 и 18
0,006
0,612
APOH_ATVVYQGER_vs_TENX_LSQLSVTDVTTSSLR
48 и 142
0,013
0,602
ITIH4_NPLVWVHASPEHVVVTR_vs_PRG2_WNFAYWAAHQPWSR
113 и 129
0,007
0,611
B2MG_VEHSDLSFSK_vs_PSG3_VSAPSGTGHLPGLNPL
50 и 134
0,011
0,604
ITIH4_NPLVWVHASPEHVVVTR_vs_PSG3_VSAPSGTGHLPGLNPL
113 и 134
0,003
0,623
B2MG_VNHVTLSQPK_vs_PSG3_VSAPSGTGHLPGLNPL
51 и 134
0,002
0,623
ITIH4_NPLVWVHASPEHVVVTR_vs_SHBG_IALGGLLFPASNLR
113 и 18
0,014
0,601
BGH3_LTLLAPLNSVFK_vs_PSG3_VSAPSGTGHLPGLNPL
52 и 134
0,007
0,610
ITIH4_NPLVWVHASPEHVVVTR_vs_TENX_LNWEAPPGAFDSFLLR
113 и 141
0,014
0,601
C1QB_VPGLYYFTYHASSR_vs_CHL1_VIAVNEVGR
55 и 66
0,005
0,613
ITIH4_QLGLPGPPDVPDHAAYHPF_vs_PSG3_VSAPSGTGHLPGLNPL
114 и 134
0,014
0,601
C1QB_VPGLYYFTYHASSR_vs_CSH_AHQLAIDTYQEFEETYIPK
55 и 80
0,005
0,614
KNG1_DIPTNSPELEETLTHTITK_vs_PSG3_VSAPSGTGHLPGLNPL
116 и 134
0,000
0,643
C1QB_VPGLYYFTYHASSR_vs_FBLN1_TGYYFDGISR
55 и 86
0,001
0,630
KNG1_DIPTNSPELEETLTHTITK_vs_SHBG_IALGGLLFPASNLR
116 и 18
0,008
0,608
C1QB_VPGLYYFTYHASSR_vs_IGF2_GIVEECCFR
55 и 103
0,010
0,605
KNG1_DIPTNSPELEETLTHTITK_vs_TENX_LNWEAPPGAFDSFLLR
116 и 141
0,010
0,606
C1QB_VPGLYYFTYHASSR_vs_PGRP2_AGLLRPDYALLGHR
55 и 126
0,010
0,605
KNG1_QVVAGLNFR_vs_FBLN1_TGYYFDGISR
117 и 86
0,013
0,601
C1QB_VPGLYYFTYHASSR_vs_PRG2_WNFAYWAAHQPWSR
55 и 129
0,001
0,632
KNG1_QVVAGLNFR_vs_PSG3_VSAPSGTGHLPGLNPL
117 и 134
0,001
0,636
C1QB_VPGLYYFTYHASSR_vs_PSG3_VSAPSGTGHLPGLNPL
55 и 134
0,000
0,649
KNG1_QVVAGLNFR_vs_SHBG_IALGGLLFPASNLR
117 и 18
0,008
0,608
C1QB_VPGLYYFTYHASSR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
55 и 135
0,013
0,602
LBP_ITGFLKPGK_vs_CHL1_VIAVNEVGR
118 и 66
0,009
0,607
C1QB_VPGLYYFTYHASSR_vs_SHBG_IALGGLLFPASNLR
55 и 18
0,001
0,641
LBP_ITGFLKPGK_vs_CRIS3_AVSPPAR
118 и 78
0,011
0,603
C1QB_VPGLYYFTYHASSR_vs_SPRL1_VLTHSELAPLR
55 и 140
0,011
0,603
LBP_ITGFLKPGK_vs_CRIS3_YEDLYSNCK
118 и 79
0,006
0,612
C1QB_VPGLYYFTYHASSR_vs_TENX_LNWEAPPGAFDSFLLR
55 и 141
0,002
0,624
LBP_ITGFLKPGK_vs_LYAM1_SYYWIGIR
118 и 120
0,011
0,604
C1QB_VPGLYYFTYHASSR_vs_TENX_LSQLSVTDVTTSSLR
55 и 142
0,003
0,619
LBP_ITGFLKPGK_vs_PRG2_WNFAYWAAHQPWSR
118 и 129
0,007
0,611
C1QB_VPGLYYFTYHASSR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
55 и 144
0,007
0,610
LBP_ITGFLKPGK_vs_PSG3_VSAPSGTGHLPGLNPL
118 и 134
0,000
0,659
CD14_LTVGAAQVPAQLLVGALR_vs_FBLN1_TGYYFDGISR
61 и 86
0,011
0,604
LBP_ITGFLKPGK_vs_SHBG_IALGGLLFPASNLR
118 и 18
0,004
0,617
CD14_LTVGAAQVPAQLLVGALR_vs_PRG2_WNFAYWAAHQPWSR
61 и 129
0,013
0,602
LBP_ITGFLKPGK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
118 и 144
0,013
0,601
CD14_LTVGAAQVPAQLLVGALR_vs_PSG3_VSAPSGTGHLPGLNPL
61 и 134
0,000
0,646
LBP_ITLPDFTGDLR_vs_C163A_INPASLDK
119 и 54
0,006
0,611
CD14_LTVGAAQVPAQLLVGALR_vs_SHBG_IALGGLLFPASNLR
61 и 18
0,006
0,611
LBP_ITLPDFTGDLR_vs_CHL1_VIAVNEVGR
119 и 66
0,003
0,621
CD14_LTVGAAQVPAQLLVGALR_vs_TENX_LNWEAPPGAFDSFLLR
61 и 141
0,004
0,616
LBP_ITLPDFTGDLR_vs_CRIS3_AVSPPAR
119 и 78
0,004
0,619
CD14_LTVGAAQVPAQLLVGALR_vs_TENX_LSQLSVTDVTTSSLR
61 и 142
0,007
0,609
LBP_ITLPDFTGDLR_vs_CRIS3_YEDLYSNCK
119 и 79
0,002
0,624
CD14_LTVGAAQVPAQLLVGALR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
61 и 144
0,011
0,604
LBP_ITLPDFTGDLR_vs_CSH_AHQLAIDTYQEFEETYIPK
119 и 80
0,013
0,601
CD14_SWLAELQQWLKPGLK_vs_FBLN1_TGYYFDGISR
62 и 86
0,013
0,602
LBP_ITLPDFTGDLR_vs_FBLN1_TGYYFDGISR
119 и 86
0,007
0,610
CD14_SWLAELQQWLKPGLK_vs_PRG2_WNFAYWAAHQPWSR
62 и 129
0,013
0,602
LBP_ITLPDFTGDLR_vs_LYAM1_SYYWIGIR
119 и 120
0,004
0,618
CD14_SWLAELQQWLKPGLK_vs_PSG3_VSAPSGTGHLPGLNPL
62 и 134
0,001
0,637
LBP_ITLPDFTGDLR_vs_PRG2_WNFAYWAAHQPWSR
119 и 129
0,003
0,623
CD14_SWLAELQQWLKPGLK_vs_SHBG_IALGGLLFPASNLR
62 и 18
0,007
0,610
LBP_ITLPDFTGDLR_vs_PSG3_VSAPSGTGHLPGLNPL
119 и 134
0,000
0,670
CD14_SWLAELQQWLKPGLK_vs_TENX_LNWEAPPGAFDSFLLR
62 и 141
0,006
0,611
LBP_ITLPDFTGDLR_vs_SHBG_IALGGLLFPASNLR
119 и 18
0,001
0,633
CD14_SWLAELQQWLKPGLK_vs_TENX_LSQLSVTDVTTSSLR
62 и 142
0,012
0,603
LBP_ITLPDFTGDLR_vs_TENX_LNWEAPPGAFDSFLLR
119 и 141
0,007
0,610
CD14_SWLAELQQWLKPGLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
62 и 144
0,014
0,601
LBP_ITLPDFTGDLR_vs_TENX_LSQLSVTDVTTSSLR
119 и 142
0,009
0,607
CFAB_YGLVTYATYPK_vs_PRG2_WNFAYWAAHQPWSR
64 и 129
0,013
0,601
LBP_ITLPDFTGDLR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
119 и 144
0,006
0,613
CFAB_YGLVTYATYPK_vs_PSG3_VSAPSGTGHLPGLNPL
64 и 134
0,000
0,646
LBP_ITLPDFTGDLR_vs_VTDB_ELPEHTVK
119 и 147
0,004
0,618
CFAB_YGLVTYATYPK_vs_SHBG_IALGGLLFPASNLR
64 и 18
0,008
0,607
PEDF_LQSLFDSPDFSK_vs_CRIS3_AVSPPAR
124 и 78
0,012
0,602
CFAB_YGLVTYATYPK_vs_TENX_LNWEAPPGAFDSFLLR
64 и 141
0,010
0,605
PEDF_LQSLFDSPDFSK_vs_CRIS3_YEDLYSNCK
124 и 79
0,008
0,608
CFAB_YGLVTYATYPK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
64 и 144
0,004
0,618
PEDF_LQSLFDSPDFSK_vs_FBLN1_TGYYFDGISR
124 и 86
0,013
0,601
CLUS_ASSIIDELFQDR_vs_PSG3_VSAPSGTGHLPGLNPL
67 и 134
0,002
0,628
PEDF_LQSLFDSPDFSK_vs_PSG3_VSAPSGTGHLPGLNPL
124 и 134
0,000
0,646
CLUS_ASSIIDELFQDR_vs_TENX_LNWEAPPGAFDSFLLR
67 и 141
0,014
0,600
PEDF_LQSLFDSPDFSK_vs_SHBG_IALGGLLFPASNLR
124 и 18
0,012
0,602
CLUS_LFDSDPITVTVPVEVSR_vs_PSG3_VSAPSGTGHLPGLNPL
68 и 134
0,004
0,617
PEDF_LQSLFDSPDFSK_vs_TENX_LNWEAPPGAFDSFLLR
124 и 141
0,004
0,619
CO5_TLLPVSKPEIR_vs_PSG3_VSAPSGTGHLPGLNPL
70 и 134
0,001
0,631
PEDF_LQSLFDSPDFSK_vs_TENX_LSQLSVTDVTTSSLR
124 и 142
0,004
0,616
CO5_TLLPVSKPEIR_vs_SHBG_IALGGLLFPASNLR
70 и 18
0,014
0,600
PEDF_TVQAVLTVPK_vs_CHL1_VIAVNEVGR
125 и 66
0,014
0,600
CO5_TLLPVSKPEIR_vs_TENX_LNWEAPPGAFDSFLLR
70 и 141
0,014
0,600
PEDF_TVQAVLTVPK_vs_FBLN1_TGYYFDGISR
125 и 86
0,008
0,608
CO5_VFQFLEK_vs_FBLN1_TGYYFDGISR
71 и 86
0,012
0,603
PEDF_TVQAVLTVPK_vs_PRG2_WNFAYWAAHQPWSR
125 и 129
0,010
0,605
CO5_VFQFLEK_vs_PSG3_VSAPSGTGHLPGLNPL
71 и 134
0,002
0,629
PEDF_TVQAVLTVPK_vs_PSG3_VSAPSGTGHLPGLNPL
125 и 134
0,000
0,649
CO5_VFQFLEK_vs_SHBG_IALGGLLFPASNLR
71 и 18
0,009
0,606
PEDF_TVQAVLTVPK_vs_SHBG_IALGGLLFPASNLR
125 и 18
0,014
0,600
CO5_VFQFLEK_vs_TENX_LNWEAPPGAFDSFLLR
71 и 141
0,011
0,604
PEDF_TVQAVLTVPK_vs_TENX_LNWEAPPGAFDSFLLR
125 и 141
0,004
0,618
CO6_ALNHLPLEYNSALYSR_vs_PSG3_VSAPSGTGHLPGLNPL
72 и 134
0,002
0,624
PEDF_TVQAVLTVPK_vs_TENX_LSQLSVTDVTTSSLR
125 и 142
0,007
0,609
CO6_ALNHLPLEYNSALYSR_vs_TENX_LNWEAPPGAFDSFLLR
72 и 141
0,013
0,602
PTGDS_GPGEDFR_vs_PSG3_VSAPSGTGHLPGLNPL
137 и 134
0,005
0,614
CO8A_SLLQPNK_vs_CHL1_VIAVNEVGR
74 и 66
0,011
0,604
THBG_AVLHIGEK_vs_PSG3_VSAPSGTGHLPGLNPL
143 и 134
0,008
0,609
CO8A_SLLQPNK_vs_FBLN1_TGYYFDGISR
74 и 86
0,013
0,601
VTNC_GQYCYELDEK_vs_CHL1_VIAVNEVGR
149 и 66
0,008
0,609
CO8A_SLLQPNK_vs_PSG3_VSAPSGTGHLPGLNPL
74 и 134
0,000
0,643
VTNC_GQYCYELDEK_vs_FBLN1_TGYYFDGISR
149 и 86
0,003
0,620
CO8A_SLLQPNK_vs_SHBG_IALGGLLFPASNLR
74 и 18
0,006
0,612
VTNC_GQYCYELDEK_vs_IGF2_GIVEECCFR
149 и 103
0,010
0,605
CO8A_SLLQPNK_vs_TENX_LNWEAPPGAFDSFLLR
74 и 141
0,002
0,624
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,005
0,614
CO8A_SLLQPNK_vs_TENX_LSQLSVTDVTTSSLR
74 и 142
0,007
0,610
VTNC_GQYCYELDEK_vs_PRG2_WNFAYWAAHQPWSR
149 и 129
0,003
0,623
CO8A_SLLQPNK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
74 и 144
0,003
0,622
VTNC_GQYCYELDEK_vs_PSG3_VSAPSGTGHLPGLNPL
149 и 134
0,000
0,669
CO8B_QALEEFQK_vs_FBLN1_TGYYFDGISR
76 и 86
0,014
0,600
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,000
0,642
CO8B_QALEEFQK_vs_PSG3_VSAPSGTGHLPGLNPL
76 и 134
0,000
0,645
VTNC_GQYCYELDEK_vs_TENX_LNWEAPPGAFDSFLLR
149 и 141
0,004
0,618
CO8B_QALEEFQK_vs_SHBG_IALGGLLFPASNLR
76 и 18
0,006
0,612
VTNC_GQYCYELDEK_vs_TENX_LSQLSVTDVTTSSLR
149 и 142
0,006
0,612
CO8B_QALEEFQK_vs_TENX_LNWEAPPGAFDSFLLR
76 и 141
0,002
0,628
VTNC_GQYCYELDEK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
149 и 144
0,002
0,624
CO8B_QALEEFQK_vs_TENX_LSQLSVTDVTTSSLR
76 и 142
0,004
0,619
VTNC_GQYCYELDEK_vs_VTDB_ELPEHTVK
149 и 147
0,003
0,623
CO8B_QALEEFQK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
76 и 144
0,002
0,627
VTNC_VDTVDPPYPR_vs_CHL1_VIAVNEVGR
150 и 66
0,010
0,605
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PSG3_VSAPSGTGHLPGLNPL
82 и 134
0,012
0,602
VTNC_VDTVDPPYPR_vs_FBLN1_TGYYFDGISR
150 и 86
0,007
0,610
ENPP2_TYLHTYESEI_vs_PSG3_VSAPSGTGHLPGLNPL
83 и 134
0,013
0,601
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,004
0,618
F13B_GDTYPAELYITGSILR_vs_PSG3_VSAPSGTGHLPGLNPL
84 и 134
0,005
0,615
VTNC_VDTVDPPYPR_vs_PRG2_WNFAYWAAHQPWSR
150 и 129
0,005
0,615
FBLN3_IPSNPSHR_vs_SHBG_IALGGLLFPASNLR
87 и 18
0,007
0,611
VTNC_VDTVDPPYPR_vs_PSG3_VSAPSGTGHLPGLNPL
150 и 134
0,000
0,669
FETUA_FSVVYAK_vs_PSG3_VSAPSGTGHLPGLNPL
88 и 134
0,001
0,633
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,001
0,634
FETUA_FSVVYAK_vs_TENX_LNWEAPPGAFDSFLLR
88 и 141
0,013
0,601
VTNC_VDTVDPPYPR_vs_TENX_LNWEAPPGAFDSFLLR
150 и 141
0,005
0,614
FETUA_HTLNQIDEVK_vs_PRG2_WNFAYWAAHQPWSR
89 и 129
0,010
0,604
VTNC_VDTVDPPYPR_vs_TENX_LSQLSVTDVTTSSLR
150 и 142
0,010
0,605
FETUA_HTLNQIDEVK_vs_PSG3_VSAPSGTGHLPGLNPL
89 и 134
0,001
0,640
VTNC_VDTVDPPYPR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
150 и 144
0,004
0,617
FETUA_HTLNQIDEVK_vs_SHBG_IALGGLLFPASNLR
89 и 18
0,013
0,601
VTNC_VDTVDPPYPR_vs_VTDB_ELPEHTVK
150 и 147
0,001
0,635
FETUA_HTLNQIDEVK_vs_TENX_LNWEAPPGAFDSFLLR
89 и 141
0,005
0,614
Таблица 52: эффективность классификации по обращениям, недели 18, 19 и 20.
AUROC обращения для недель беременности от 18 0/7 до 20 6/7 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, с BMI-стратификацией (>22≤37).
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
A2GL_DLLLPQPDLR_vs_CRIS3_AVSPPAR
34 и 78
0,015
0,621
CO8B_QALEEFQK_vs_PSG3_VSAPSGTGHLPGLNPL
76 и 134
0,003
0,648
A2GL_DLLLPQPDLR_vs_CRIS3_YEDLYSNCK
34 и 79
0,009
0,631
CO8B_QALEEFQK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
76 и 135
0,016
0,620
A2GL_DLLLPQPDLR_vs_LYAM1_SYYWIGIR
34 и 120
0,006
0,638
CO8B_QALEEFQK_vs_PSG9_LFIPQITR
76 и 136
0,032
0,607
A2GL_DLLLPQPDLR_vs_PGRP2_AGLLRPDYALLGHR
34 и 126
0,032
0,608
CO8B_QALEEFQK_vs_SHBG_IALGGLLFPASNLR
76 и 18
0,030
0,609
A2GL_DLLLPQPDLR_vs_PSG3_VSAPSGTGHLPGLNPL
34 и 134
0,003
0,648
CO8B_QALEEFQK_vs_SPRL1_VLTHSELAPLR
76 и 140
0,009
0,631
A2GL_DLLLPQPDLR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
34 и 135
0,014
0,623
CO8B_QALEEFQK_vs_TENX_LNWEAPPGAFDSFLLR
76 и 141
0,002
0,659
A2GL_DLLLPQPDLR_vs_PSG9_LFIPQITR
34 и 136
0,034
0,606
CO8B_QALEEFQK_vs_TENX_LSQLSVTDVTTSSLR
76 и 142
0,003
0,651
A2GL_DLLLPQPDLR_vs_SHBG_IALGGLLFPASNLR
34 и 18
0,010
0,628
CO8B_QALEEFQK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
76 и 144
0,006
0,639
A2GL_DLLLPQPDLR_vs_SPRL1_VLTHSELAPLR
34 и 140
0,017
0,620
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_LYAM1_SYYWIGIR
82 и 120
0,041
0,602
A2GL_DLLLPQPDLR_vs_TENX_LNWEAPPGAFDSFLLR
34 и 141
0,031
0,608
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_SHBG_IALGGLLFPASNLR
82 и 18
0,038
0,604
A2GL_DLLLPQPDLR_vs_TENX_LSQLSVTDVTTSSLR
34 и 142
0,042
0,602
F13B_GDTYPAELYITGSILR_vs_CRIS3_YEDLYSNCK
84 и 79
0,021
0,615
A2GL_DLLLPQPDLR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
34 и 144
0,020
0,616
F13B_GDTYPAELYITGSILR_vs_PSG3_VSAPSGTGHLPGLNPL
84 и 134
0,019
0,617
AFAM_DADPDTFFAK_vs_CRIS3_YEDLYSNCK
37 и 79
0,018
0,619
F13B_GDTYPAELYITGSILR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
84 и 135
0,037
0,604
AFAM_DADPDTFFAK_vs_IBP3_FLNVLSPR
37 и 99
0,041
0,602
F13B_GDTYPAELYITGSILR_vs_TENX_LNWEAPPGAFDSFLLR
84 и 141
0,041
0,602
AFAM_DADPDTFFAK_vs_IBP3_YGQPLPGYTTK
37 и 100
0,036
0,605
F13B_GDTYPAELYITGSILR_vs_TENX_LSQLSVTDVTTSSLR
84 и 142
0,039
0,603
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,013
0,624
FBLN3_IPSNPSHR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
87 и 135
0,022
0,615
AFAM_DADPDTFFAK_vs_LYAM1_SYYWIGIR
37 и 120
0,029
0,609
FBLN3_IPSNPSHR_vs_SHBG_IALGGLLFPASNLR
87 и 18
0,038
0,604
AFAM_DADPDTFFAK_vs_PSG3_VSAPSGTGHLPGLNPL
37 и 134
0,003
0,650
FETUA_FSVVYAK_vs_PSG3_VSAPSGTGHLPGLNPL
88 и 134
0,029
0,609
AFAM_DADPDTFFAK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
37 и 135
0,019
0,618
FETUA_HTLNQIDEVK_vs_PSG3_VSAPSGTGHLPGLNPL
89 и 134
0,023
0,614
AFAM_DADPDTFFAK_vs_PSG9_LFIPQITR
37 и 136
0,042
0,602
FETUA_HTLNQIDEVK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
89 и 135
0,038
0,604
AFAM_DADPDTFFAK_vs_SPRL1_VLTHSELAPLR
37 и 140
0,042
0,602
FETUA_HTLNQIDEVK_vs_TENX_LNWEAPPGAFDSFLLR
89 и 141
0,024
0,613
AFAM_DADPDTFFAK_vs_TENX_LNWEAPPGAFDSFLLR
37 и 141
0,011
0,627
HABP2_FLNWIK_vs_CHL1_VIAVNEVGR
92 и 66
0,015
0,622
AFAM_DADPDTFFAK_vs_TENX_LSQLSVTDVTTSSLR
37 и 142
0,022
0,614
HABP2_FLNWIK_vs_CRIS3_AVSPPAR
92 и 78
0,028
0,610
AFAM_DADPDTFFAK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
37 и 144
0,018
0,619
HABP2_FLNWIK_vs_CRIS3_YEDLYSNCK
92 и 79
0,019
0,618
AFAM_HFQNLGK_vs_CHL1_VIAVNEVGR
38 и 66
0,018
0,618
HABP2_FLNWIK_vs_IGF2_GIVEECCFR
92 и 103
0,014
0,623
AFAM_HFQNLGK_vs_CRIS3_YEDLYSNCK
38 и 79
0,021
0,615
HABP2_FLNWIK_vs_LYAM1_SYYWIGIR
92 и 120
0,010
0,629
AFAM_HFQNLGK_vs_IBP3_FLNVLSPR
38 и 99
0,033
0,607
HABP2_FLNWIK_vs_NCAM1_GLGEISAASEFK
92 и 121
0,026
0,612
AFAM_HFQNLGK_vs_IBP3_YGQPLPGYTTK
38 и 100
0,026
0,611
HABP2_FLNWIK_vs_PGRP2_AGLLRPDYALLGHR
92 и 126
0,044
0,601
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,024
0,613
HABP2_FLNWIK_vs_PSG3_VSAPSGTGHLPGLNPL
92 и 134
0,003
0,648
AFAM_HFQNLGK_vs_LYAM1_SYYWIGIR
38 и 120
0,023
0,614
HABP2_FLNWIK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
92 и 135
0,015
0,622
AFAM_HFQNLGK_vs_PSG3_VSAPSGTGHLPGLNPL
38 и 134
0,003
0,648
HABP2_FLNWIK_vs_PSG9_LFIPQITR
92 и 136
0,033
0,607
AFAM_HFQNLGK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
38 и 135
0,012
0,626
HABP2_FLNWIK_vs_SHBG_IALGGLLFPASNLR
92 и 18
0,022
0,615
AFAM_HFQNLGK_vs_PSG9_LFIPQITR
38 и 136
0,029
0,610
HABP2_FLNWIK_vs_SPRL1_VLTHSELAPLR
92 и 140
0,015
0,622
AFAM_HFQNLGK_vs_SHBG_IALGGLLFPASNLR
38 и 18
0,046
0,600
HABP2_FLNWIK_vs_TENX_LNWEAPPGAFDSFLLR
92 и 141
0,007
0,636
AFAM_HFQNLGK_vs_SPRL1_VLTHSELAPLR
38 и 140
0,017
0,619
HABP2_FLNWIK_vs_TENX_LSQLSVTDVTTSSLR
92 и 142
0,008
0,632
AFAM_HFQNLGK_vs_TENX_LNWEAPPGAFDSFLLR
38 и 141
0,009
0,632
HABP2_FLNWIK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
92 и 144
0,016
0,620
AFAM_HFQNLGK_vs_TENX_LSQLSVTDVTTSSLR
38 и 142
0,015
0,622
HABP2_FLNWIK_vs_VTDB_ELPEHTVK
92 и 147
0,019
0,618
AFAM_HFQNLGK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
38 и 144
0,029
0,610
HEMO_NFPSPVDAAFR_vs_CRIS3_YEDLYSNCK
93 и 79
0,036
0,605
AFAM_HFQNLGK_vs_VTDB_ELPEHTVK
38 и 147
0,037
0,605
HEMO_NFPSPVDAAFR_vs_LYAM1_SYYWIGIR
93 и 120
0,041
0,603
ANGT_DPTFIPAPIQAK_vs_CHL1_VIAVNEVGR
42 и 66
0,010
0,630
HEMO_NFPSPVDAAFR_vs_PSG3_VSAPSGTGHLPGLNPL
93 и 134
0,036
0,605
ANGT_DPTFIPAPIQAK_vs_CRIS3_AVSPPAR
42 и 78
0,010
0,628
HEMO_NFPSPVDAAFR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
93 и 135
0,030
0,609
ANGT_DPTFIPAPIQAK_vs_CRIS3_YEDLYSNCK
42 и 79
0,006
0,637
HEMO_NFPSPVDAAFR_vs_TENX_LNWEAPPGAFDSFLLR
93 и 141
0,013
0,624
ANGT_DPTFIPAPIQAK_vs_CSH_AHQLAIDTYQEFEETYIPK
42 и 80
0,042
0,602
HEMO_NFPSPVDAAFR_vs_TENX_LSQLSVTDVTTSSLR
93 и 142
0,037
0,604
ANGT_DPTFIPAPIQAK_vs_FBLN1_TGYYFDGISR
42 и 86
0,036
0,605
HLACI_WAAVVVPSGEEQR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
95 и 135
0,043
0,602
ANGT_DPTFIPAPIQAK_vs_LYAM1_SYYWIGIR
42 и 120
0,011
0,627
IBP4_QCHPALDGQR_vs_CHL1_VIAVNEVGR
2 и 66
0,014
0,624
ANGT_DPTFIPAPIQAK_vs_NCAM1_GLGEISAASEFK
42 и 121
0,025
0,613
IBP4_QCHPALDGQR_vs_CRIS3_AVSPPAR
2 и 78
0,006
0,639
ANGT_DPTFIPAPIQAK_vs_PGRP2_AGLLRPDYALLGHR
42 и 126
0,028
0,610
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,002
0,652
ANGT_DPTFIPAPIQAK_vs_PSG3_VSAPSGTGHLPGLNPL
42 и 134
0,001
0,669
IBP4_QCHPALDGQR_vs_IGF2_GIVEECCFR
2 и 103
0,018
0,619
ANGT_DPTFIPAPIQAK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
42 и 135
0,013
0,625
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,003
0,648
ANGT_DPTFIPAPIQAK_vs_PSG9_LFIPQITR
42 и 136
0,031
0,608
IBP4_QCHPALDGQR_vs_NCAM1_GLGEISAASEFK
2 и 121
0,041
0,602
ANGT_DPTFIPAPIQAK_vs_SHBG_IALGGLLFPASNLR
42 и 18
0,002
0,655
IBP4_QCHPALDGQR_vs_PGRP2_AGLLRPDYALLGHR
2 и 126
0,010
0,630
ANGT_DPTFIPAPIQAK_vs_SOM2.CSH_SVEGSCGF
42 и 139
0,019
0,619
IBP4_QCHPALDGQR_vs_PSG3_VSAPSGTGHLPGLNPL
2 и 134
0,000
0,675
ANGT_DPTFIPAPIQAK_vs_SPRL1_VLTHSELAPLR
42 и 140
0,019
0,618
IBP4_QCHPALDGQR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
2 и 135
0,010
0,630
ANGT_DPTFIPAPIQAK_vs_TENX_LNWEAPPGAFDSFLLR
42 и 141
0,010
0,628
IBP4_QCHPALDGQR_vs_PSG9_LFIPQITR
2 и 136
0,023
0,614
ANGT_DPTFIPAPIQAK_vs_TENX_LSQLSVTDVTTSSLR
42 и 142
0,011
0,627
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,004
0,643
ANGT_DPTFIPAPIQAK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
42 и 144
0,019
0,617
IBP4_QCHPALDGQR_vs_SPRL1_VLTHSELAPLR
2 и 140
0,007
0,635
ANGT_DPTFIPAPIQAK_vs_VTDB_ELPEHTVK
42 и 147
0,032
0,607
IBP4_QCHPALDGQR_vs_TENX_LNWEAPPGAFDSFLLR
2 и 141
0,003
0,648
APOC3_GWVTDGFSSLK_vs_ALS_IRPHTFTGLSGLR
47 и 40
0,013
0,624
IBP4_QCHPALDGQR_vs_TENX_LSQLSVTDVTTSSLR
2 и 142
0,008
0,633
APOC3_GWVTDGFSSLK_vs_C163A_INPASLDK
47 и 54
0,001
0,662
IBP4_QCHPALDGQR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
2 и 144
0,006
0,638
APOC3_GWVTDGFSSLK_vs_CHL1_VIAVNEVGR
47 и 66
0,013
0,624
IBP4_QCHPALDGQR_vs_VTDB_ELPEHTVK
2 и 147
0,026
0,611
APOC3_GWVTDGFSSLK_vs_CRIS3_AVSPPAR
47 и 78
0,001
0,663
INHBC_LDFHFSSDR_vs_ALS_IRPHTFTGLSGLR
107 и 40
0,023
0,614
APOC3_GWVTDGFSSLK_vs_CRIS3_YEDLYSNCK
47 и 79
0,001
0,669
INHBC_LDFHFSSDR_vs_C163A_INPASLDK
107 и 54
0,030
0,609
APOC3_GWVTDGFSSLK_vs_CSH_AHQLAIDTYQEFEETYIPK
47 и 80
0,016
0,621
INHBC_LDFHFSSDR_vs_CHL1_VIAVNEVGR
107 и 66
0,008
0,633
APOC3_GWVTDGFSSLK_vs_CSH_ISLLLIESWLEPVR
47 и 81
0,024
0,613
INHBC_LDFHFSSDR_vs_CRIS3_AVSPPAR
107 и 78
0,019
0,618
APOC3_GWVTDGFSSLK_vs_FBLN1_TGYYFDGISR
47 и 86
0,015
0,622
INHBC_LDFHFSSDR_vs_CRIS3_YEDLYSNCK
107 и 79
0,010
0,629
APOC3_GWVTDGFSSLK_vs_IBP2_LIQGAPTIR
47 и 98
0,035
0,606
INHBC_LDFHFSSDR_vs_FBLN1_TGYYFDGISR
107 и 86
0,029
0,610
APOC3_GWVTDGFSSLK_vs_IBP3_FLNVLSPR
47 и 99
0,004
0,643
INHBC_LDFHFSSDR_vs_IBP3_FLNVLSPR
107 и 99
0,007
0,636
APOC3_GWVTDGFSSLK_vs_IBP3_YGQPLPGYTTK
47 и 100
0,008
0,633
INHBC_LDFHFSSDR_vs_IBP3_YGQPLPGYTTK
107 и 100
0,007
0,635
APOC3_GWVTDGFSSLK_vs_IGF2_GIVEECCFR
47 и 103
0,005
0,641
INHBC_LDFHFSSDR_vs_IGF2_GIVEECCFR
107 и 103
0,003
0,648
APOC3_GWVTDGFSSLK_vs_ITIH4_ILDDLSPR
47 и 112
0,016
0,620
INHBC_LDFHFSSDR_vs_ITIH4_ILDDLSPR
107 и 112
0,024
0,613
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,001
0,660
INHBC_LDFHFSSDR_vs_LYAM1_SYYWIGIR
107 и 120
0,016
0,621
APOC3_GWVTDGFSSLK_vs_NCAM1_GLGEISAASEFK
47 и 121
0,014
0,623
INHBC_LDFHFSSDR_vs_NCAM1_GLGEISAASEFK
107 и 121
0,018
0,618
APOC3_GWVTDGFSSLK_vs_PGRP2_AGLLRPDYALLGHR
47 и 126
0,008
0,633
INHBC_LDFHFSSDR_vs_PGRP2_AGLLRPDYALLGHR
107 и 126
0,011
0,628
APOC3_GWVTDGFSSLK_vs_PRG2_WNFAYWAAHQPWSR
47 и 129
0,041
0,603
INHBC_LDFHFSSDR_vs_PSG3_VSAPSGTGHLPGLNPL
107 и 134
0,000
0,681
APOC3_GWVTDGFSSLK_vs_PSG1_FQLPGQK
47 и 131
0,022
0,615
INHBC_LDFHFSSDR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
107 и 135
0,012
0,626
APOC3_GWVTDGFSSLK_vs_PSG3_VSAPSGTGHLPGLNPL
47 и 134
0,001
0,664
INHBC_LDFHFSSDR_vs_PSG9_LFIPQITR
107 и 136
0,015
0,622
APOC3_GWVTDGFSSLK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
47 и 135
0,005
0,642
INHBC_LDFHFSSDR_vs_SHBG_IALGGLLFPASNLR
107 и 18
0,007
0,634
APOC3_GWVTDGFSSLK_vs_PSG9_LFIPQITR
47 и 136
0,007
0,636
INHBC_LDFHFSSDR_vs_SPRL1_VLTHSELAPLR
107 и 140
0,003
0,647
APOC3_GWVTDGFSSLK_vs_SHBG_IALGGLLFPASNLR
47 и 18
0,008
0,633
INHBC_LDFHFSSDR_vs_TENX_LNWEAPPGAFDSFLLR
107 и 141
0,000
0,675
APOC3_GWVTDGFSSLK_vs_SOM2.CSH_NYGLLYCFR
47 и 138
0,035
0,606
INHBC_LDFHFSSDR_vs_TENX_LSQLSVTDVTTSSLR
107 и 142
0,001
0,664
APOC3_GWVTDGFSSLK_vs_SOM2.CSH_SVEGSCGF
47 и 139
0,015
0,624
INHBC_LDFHFSSDR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
107 и 144
0,006
0,639
APOC3_GWVTDGFSSLK_vs_SPRL1_VLTHSELAPLR
47 и 140
0,010
0,629
INHBC_LDFHFSSDR_vs_VTDB_ELPEHTVK
107 и 147
0,005
0,639
APOC3_GWVTDGFSSLK_vs_TENX_LNWEAPPGAFDSFLLR
47 и 141
0,005
0,641
ITIH3_ALDLSLK_vs_CRIS3_AVSPPAR
111 и 78
0,040
0,603
APOC3_GWVTDGFSSLK_vs_TENX_LSQLSVTDVTTSSLR
47 и 142
0,007
0,636
ITIH3_ALDLSLK_vs_CRIS3_YEDLYSNCK
111 и 79
0,023
0,614
APOC3_GWVTDGFSSLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
47 и 144
0,004
0,644
ITIH3_ALDLSLK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
111 и 135
0,037
0,605
APOC3_GWVTDGFSSLK_vs_VTDB_ELPEHTVK
47 и 147
0,007
0,634
ITIH3_ALDLSLK_vs_SHBG_IALGGLLFPASNLR
111 и 18
0,036
0,605
APOH_ATVVYQGER_vs_CRIS3_YEDLYSNCK
48 и 79
0,025
0,612
ITIH4_NPLVWVHASPEHVVVTR_vs_CRIS3_AVSPPAR
113 и 78
0,016
0,621
APOH_ATVVYQGER_vs_LYAM1_SYYWIGIR
48 и 120
0,031
0,608
ITIH4_NPLVWVHASPEHVVVTR_vs_CRIS3_YEDLYSNCK
113 и 79
0,011
0,628
APOH_ATVVYQGER_vs_PSG3_VSAPSGTGHLPGLNPL
48 и 134
0,013
0,625
ITIH4_NPLVWVHASPEHVVVTR_vs_LYAM1_SYYWIGIR
113 и 120
0,009
0,631
APOH_ATVVYQGER_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
48 и 135
0,008
0,633
ITIH4_NPLVWVHASPEHVVVTR_vs_PSG3_VSAPSGTGHLPGLNPL
113 и 134
0,020
0,617
APOH_ATVVYQGER_vs_PSG9_LFIPQITR
48 и 136
0,020
0,617
ITIH4_NPLVWVHASPEHVVVTR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
113 и 135
0,010
0,630
APOH_ATVVYQGER_vs_SPRL1_VLTHSELAPLR
48 и 140
0,036
0,605
ITIH4_NPLVWVHASPEHVVVTR_vs_PSG9_LFIPQITR
113 и 136
0,031
0,608
APOH_ATVVYQGER_vs_TENX_LNWEAPPGAFDSFLLR
48 и 141
0,028
0,610
ITIH4_NPLVWVHASPEHVVVTR_vs_SHBG_IALGGLLFPASNLR
113 и 18
0,033
0,607
APOH_ATVVYQGER_vs_TENX_LSQLSVTDVTTSSLR
48 и 142
0,027
0,611
ITIH4_NPLVWVHASPEHVVVTR_vs_TENX_LNWEAPPGAFDSFLLR
113 и 141
0,018
0,619
B2MG_VNHVTLSQPK_vs_CRIS3_YEDLYSNCK
51 и 79
0,030
0,609
ITIH4_NPLVWVHASPEHVVVTR_vs_TENX_LSQLSVTDVTTSSLR
113 и 142
0,021
0,615
B2MG_VNHVTLSQPK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
51 и 135
0,024
0,613
ITIH4_QLGLPGPPDVPDHAAYHPF_vs_CRIS3_YEDLYSNCK
114 и 79
0,042
0,602
BGH3_LTLLAPLNSVFK_vs_CHL1_VIAVNEVGR
52 и 66
0,036
0,605
ITIH4_QLGLPGPPDVPDHAAYHPF_vs_PSG3_VSAPSGTGHLPGLNPL
114 и 134
0,035
0,606
BGH3_LTLLAPLNSVFK_vs_CRIS3_AVSPPAR
52 и 78
0,039
0,604
ITIH4_QLGLPGPPDVPDHAAYHPF_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
114 и 135
0,035
0,606
BGH3_LTLLAPLNSVFK_vs_CRIS3_YEDLYSNCK
52 и 79
0,022
0,615
KNG1_DIPTNSPELEETLTHTITK_vs_CHL1_VIAVNEVGR
116 и 66
0,045
0,601
BGH3_LTLLAPLNSVFK_vs_LYAM1_SYYWIGIR
52 и 120
0,027
0,611
KNG1_DIPTNSPELEETLTHTITK_vs_CRIS3_AVSPPAR
116 и 78
0,036
0,605
BGH3_LTLLAPLNSVFK_vs_PSG3_VSAPSGTGHLPGLNPL
52 и 134
0,028
0,610
KNG1_DIPTNSPELEETLTHTITK_vs_CRIS3_YEDLYSNCK
116 и 79
0,019
0,617
BGH3_LTLLAPLNSVFK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
52 и 135
0,030
0,609
KNG1_DIPTNSPELEETLTHTITK_vs_LYAM1_SYYWIGIR
116 и 120
0,014
0,623
BGH3_LTLLAPLNSVFK_vs_PSG9_LFIPQITR
52 и 136
0,046
0,600
KNG1_DIPTNSPELEETLTHTITK_vs_PSG3_VSAPSGTGHLPGLNPL
116 и 134
0,012
0,626
BGH3_LTLLAPLNSVFK_vs_SHBG_IALGGLLFPASNLR
52 и 18
0,043
0,602
KNG1_DIPTNSPELEETLTHTITK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
116 и 135
0,035
0,606
BGH3_LTLLAPLNSVFK_vs_TENX_LNWEAPPGAFDSFLLR
52 и 141
0,024
0,613
KNG1_DIPTNSPELEETLTHTITK_vs_TENX_LNWEAPPGAFDSFLLR
116 и 141
0,028
0,610
C1QB_VPGLYYFTYHASSR_vs_ALS_IRPHTFTGLSGLR
55 и 40
0,033
0,607
KNG1_DIPTNSPELEETLTHTITK_vs_TENX_LSQLSVTDVTTSSLR
116 и 142
0,034
0,606
C1QB_VPGLYYFTYHASSR_vs_C163A_INPASLDK
55 и 54
0,014
0,623
KNG1_QVVAGLNFR_vs_CHL1_VIAVNEVGR
117 и 66
0,040
0,603
C1QB_VPGLYYFTYHASSR_vs_CHL1_VIAVNEVGR
55 и 66
0,005
0,641
KNG1_QVVAGLNFR_vs_CRIS3_YEDLYSNCK
117 и 79
0,027
0,611
C1QB_VPGLYYFTYHASSR_vs_CRIS3_AVSPPAR
55 и 78
0,004
0,644
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,015
0,622
C1QB_VPGLYYFTYHASSR_vs_CRIS3_YEDLYSNCK
55 и 79
0,004
0,646
KNG1_QVVAGLNFR_vs_PSG3_VSAPSGTGHLPGLNPL
117 и 134
0,016
0,621
C1QB_VPGLYYFTYHASSR_vs_CSH_AHQLAIDTYQEFEETYIPK
55 и 80
0,007
0,636
KNG1_QVVAGLNFR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
117 и 135
0,028
0,610
C1QB_VPGLYYFTYHASSR_vs_CSH_ISLLLIESWLEPVR
55 и 81
0,012
0,627
LBP_ITGFLKPGK_vs_CHL1_VIAVNEVGR
118 и 66
0,046
0,600
C1QB_VPGLYYFTYHASSR_vs_FBLN1_TGYYFDGISR
55 и 86
0,008
0,634
LBP_ITGFLKPGK_vs_CRIS3_AVSPPAR
118 и 78
0,025
0,613
C1QB_VPGLYYFTYHASSR_vs_IBP2_LIQGAPTIR
55 и 98
0,031
0,608
LBP_ITGFLKPGK_vs_CRIS3_YEDLYSNCK
118 и 79
0,013
0,624
C1QB_VPGLYYFTYHASSR_vs_IBP3_FLNVLSPR
55 и 99
0,010
0,629
LBP_ITGFLKPGK_vs_LYAM1_SYYWIGIR
118 и 120
0,019
0,618
C1QB_VPGLYYFTYHASSR_vs_IBP3_YGQPLPGYTTK
55 и 100
0,012
0,626
LBP_ITGFLKPGK_vs_PSG3_VSAPSGTGHLPGLNPL
118 и 134
0,004
0,646
C1QB_VPGLYYFTYHASSR_vs_IGF2_GIVEECCFR
55 и 103
0,004
0,645
LBP_ITGFLKPGK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
118 и 135
0,033
0,607
C1QB_VPGLYYFTYHASSR_vs_ITIH4_ILDDLSPR
55 и 112
0,013
0,625
LBP_ITGFLKPGK_vs_SHBG_IALGGLLFPASNLR
118 и 18
0,029
0,610
C1QB_VPGLYYFTYHASSR_vs_LYAM1_SYYWIGIR
55 и 120
0,003
0,651
LBP_ITLPDFTGDLR_vs_C163A_INPASLDK
119 и 54
0,021
0,616
C1QB_VPGLYYFTYHASSR_vs_NCAM1_GLGEISAASEFK
55 и 121
0,008
0,633
LBP_ITLPDFTGDLR_vs_CHL1_VIAVNEVGR
119 и 66
0,014
0,623
C1QB_VPGLYYFTYHASSR_vs_PGRP2_AGLLRPDYALLGHR
55 и 126
0,002
0,652
LBP_ITLPDFTGDLR_vs_CRIS3_AVSPPAR
119 и 78
0,006
0,638
C1QB_VPGLYYFTYHASSR_vs_PRG2_WNFAYWAAHQPWSR
55 и 129
0,020
0,617
LBP_ITLPDFTGDLR_vs_CRIS3_YEDLYSNCK
119 и 79
0,004
0,644
C1QB_VPGLYYFTYHASSR_vs_PSG1_FQLPGQK
55 и 131
0,024
0,613
LBP_ITLPDFTGDLR_vs_LYAM1_SYYWIGIR
119 и 120
0,005
0,642
C1QB_VPGLYYFTYHASSR_vs_PSG3_VSAPSGTGHLPGLNPL
55 и 134
0,001
0,671
LBP_ITLPDFTGDLR_vs_NCAM1_GLGEISAASEFK
119 и 121
0,025
0,613
C1QB_VPGLYYFTYHASSR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
55 и 135
0,002
0,658
LBP_ITLPDFTGDLR_vs_PGRP2_AGLLRPDYALLGHR
119 и 126
0,028
0,610
C1QB_VPGLYYFTYHASSR_vs_PSG9_LFIPQITR
55 и 136
0,004
0,643
LBP_ITLPDFTGDLR_vs_PSG3_VSAPSGTGHLPGLNPL
119 и 134
0,001
0,665
C1QB_VPGLYYFTYHASSR_vs_SHBG_IALGGLLFPASNLR
55 и 18
0,001
0,668
LBP_ITLPDFTGDLR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
119 и 135
0,012
0,626
C1QB_VPGLYYFTYHASSR_vs_SOM2.CSH_NYGLLYCFR
55 и 138
0,014
0,624
LBP_ITLPDFTGDLR_vs_PSG9_LFIPQITR
119 и 136
0,033
0,607
C1QB_VPGLYYFTYHASSR_vs_SOM2.CSH_SVEGSCGF
55 и 139
0,009
0,631
LBP_ITLPDFTGDLR_vs_SHBG_IALGGLLFPASNLR
119 и 18
0,007
0,636
C1QB_VPGLYYFTYHASSR_vs_SPRL1_VLTHSELAPLR
55 и 140
0,003
0,648
LBP_ITLPDFTGDLR_vs_SOM2.CSH_SVEGSCGF
119 и 139
0,022
0,616
C1QB_VPGLYYFTYHASSR_vs_TENX_LNWEAPPGAFDSFLLR
55 и 141
0,002
0,654
LBP_ITLPDFTGDLR_vs_SPRL1_VLTHSELAPLR
119 и 140
0,034
0,606
C1QB_VPGLYYFTYHASSR_vs_TENX_LSQLSVTDVTTSSLR
55 и 142
0,002
0,657
LBP_ITLPDFTGDLR_vs_TENX_LNWEAPPGAFDSFLLR
119 и 141
0,027
0,611
C1QB_VPGLYYFTYHASSR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
55 и 144
0,006
0,638
LBP_ITLPDFTGDLR_vs_TENX_LSQLSVTDVTTSSLR
119 и 142
0,024
0,613
C1QB_VPGLYYFTYHASSR_vs_VTDB_ELPEHTVK
55 и 147
0,013
0,625
LBP_ITLPDFTGDLR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
119 и 144
0,027
0,611
CBPN_EALIQFLEQVHQGIK_vs_LYAM1_SYYWIGIR
59 и 120
0,025
0,613
LBP_ITLPDFTGDLR_vs_VTDB_ELPEHTVK
119 и 147
0,023
0,614
CBPN_EALIQFLEQVHQGIK_vs_PSG3_VSAPSGTGHLPGLNPL
59 и 134
0,038
0,604
PEDF_LQSLFDSPDFSK_vs_CHL1_VIAVNEVGR
124 и 66
0,038
0,604
CBPN_EALIQFLEQVHQGIK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
59 и 135
0,025
0,612
PEDF_LQSLFDSPDFSK_vs_CRIS3_AVSPPAR
124 и 78
0,012
0,626
CBPN_EALIQFLEQVHQGIK_vs_TENX_LNWEAPPGAFDSFLLR
59 и 141
0,037
0,605
PEDF_LQSLFDSPDFSK_vs_CRIS3_YEDLYSNCK
124 и 79
0,008
0,633
CBPN_NNANGVDLNR_vs_LYAM1_SYYWIGIR
60 и 120
0,039
0,603
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,015
0,622
CBPN_NNANGVDLNR_vs_PSG3_VSAPSGTGHLPGLNPL
60 и 134
0,026
0,611
PEDF_LQSLFDSPDFSK_vs_PSG3_VSAPSGTGHLPGLNPL
124 и 134
0,006
0,637
CBPN_NNANGVDLNR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
60 и 135
0,025
0,613
PEDF_LQSLFDSPDFSK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
124 и 135
0,024
0,613
CBPN_NNANGVDLNR_vs_TENX_LNWEAPPGAFDSFLLR
60 и 141
0,043
0,601
PEDF_LQSLFDSPDFSK_vs_SHBG_IALGGLLFPASNLR
124 и 18
0,043
0,601
CD14_LTVGAAQVPAQLLVGALR_vs_CRIS3_AVSPPAR
61 и 78
0,043
0,601
PEDF_LQSLFDSPDFSK_vs_TENX_LNWEAPPGAFDSFLLR
124 и 141
0,009
0,631
CD14_LTVGAAQVPAQLLVGALR_vs_CRIS3_YEDLYSNCK
61 и 79
0,020
0,617
PEDF_LQSLFDSPDFSK_vs_TENX_LSQLSVTDVTTSSLR
124 и 142
0,010
0,630
CD14_LTVGAAQVPAQLLVGALR_vs_LYAM1_SYYWIGIR
61 и 120
0,012
0,627
PEDF_TVQAVLTVPK_vs_CHL1_VIAVNEVGR
125 и 66
0,018
0,619
CD14_LTVGAAQVPAQLLVGALR_vs_PSG3_VSAPSGTGHLPGLNPL
61 и 134
0,008
0,632
PEDF_TVQAVLTVPK_vs_CRIS3_AVSPPAR
125 и 78
0,017
0,619
CD14_LTVGAAQVPAQLLVGALR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
61 и 135
0,025
0,612
PEDF_TVQAVLTVPK_vs_CRIS3_YEDLYSNCK
125 и 79
0,011
0,628
CD14_LTVGAAQVPAQLLVGALR_vs_SPRL1_VLTHSELAPLR
61 и 140
0,028
0,610
PEDF_TVQAVLTVPK_vs_LYAM1_SYYWIGIR
125 и 120
0,010
0,629
CD14_LTVGAAQVPAQLLVGALR_vs_TENX_LNWEAPPGAFDSFLLR
61 и 141
0,008
0,634
PEDF_TVQAVLTVPK_vs_PSG3_VSAPSGTGHLPGLNPL
125 и 134
0,002
0,654
CD14_LTVGAAQVPAQLLVGALR_vs_TENX_LSQLSVTDVTTSSLR
61 и 142
0,009
0,632
PEDF_TVQAVLTVPK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
125 и 135
0,021
0,616
CD14_LTVGAAQVPAQLLVGALR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
61 и 144
0,024
0,613
PEDF_TVQAVLTVPK_vs_PSG9_LFIPQITR
125 и 136
0,040
0,603
CD14_SWLAELQQWLKPGLK_vs_CRIS3_YEDLYSNCK
62 и 79
0,041
0,603
PEDF_TVQAVLTVPK_vs_SHBG_IALGGLLFPASNLR
125 и 18
0,043
0,602
CD14_SWLAELQQWLKPGLK_vs_LYAM1_SYYWIGIR
62 и 120
0,022
0,614
PEDF_TVQAVLTVPK_vs_SPRL1_VLTHSELAPLR
125 и 140
0,031
0,608
CD14_SWLAELQQWLKPGLK_vs_PSG3_VSAPSGTGHLPGLNPL
62 и 134
0,015
0,622
PEDF_TVQAVLTVPK_vs_TENX_LNWEAPPGAFDSFLLR
125 и 141
0,006
0,637
CD14_SWLAELQQWLKPGLK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
62 и 135
0,027
0,611
PEDF_TVQAVLTVPK_vs_TENX_LSQLSVTDVTTSSLR
125 и 142
0,009
0,632
CD14_SWLAELQQWLKPGLK_vs_SPRL1_VLTHSELAPLR
62 и 140
0,038
0,604
PEDF_TVQAVLTVPK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
125 и 144
0,038
0,604
CD14_SWLAELQQWLKPGLK_vs_TENX_LNWEAPPGAFDSFLLR
62 и 141
0,013
0,625
PRDX2_GLFIIDGK_vs_CRIS3_AVSPPAR
128 и 78
0,029
0,609
CD14_SWLAELQQWLKPGLK_vs_TENX_LSQLSVTDVTTSSLR
62 и 142
0,016
0,621
PRDX2_GLFIIDGK_vs_CRIS3_YEDLYSNCK
128 и 79
0,016
0,621
CD14_SWLAELQQWLKPGLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
62 и 144
0,033
0,607
PRDX2_GLFIIDGK_vs_PSG3_VSAPSGTGHLPGLNPL
128 и 134
0,014
0,623
CFAB_YGLVTYATYPK_vs_CHL1_VIAVNEVGR
64 и 66
0,033
0,607
PRDX2_GLFIIDGK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
128 и 135
0,029
0,609
CFAB_YGLVTYATYPK_vs_CRIS3_AVSPPAR
64 и 78
0,039
0,603
PRDX2_GLFIIDGK_vs_PSG9_LFIPQITR
128 и 136
0,041
0,602
CFAB_YGLVTYATYPK_vs_CRIS3_YEDLYSNCK
64 и 79
0,023
0,614
PRDX2_GLFIIDGK_vs_SHBG_IALGGLLFPASNLR
128 и 18
0,044
0,601
CFAB_YGLVTYATYPK_vs_LYAM1_SYYWIGIR
64 и 120
0,031
0,608
PRDX2_GLFIIDGK_vs_TENX_LNWEAPPGAFDSFLLR
128 и 141
0,044
0,601
CFAB_YGLVTYATYPK_vs_PSG3_VSAPSGTGHLPGLNPL
64 и 134
0,003
0,648
PSG2_IHPSYTNYR_vs_CHL1_VIAVNEVGR
133 и 66
0,037
0,605
CFAB_YGLVTYATYPK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
64 и 135
0,020
0,616
PSG2_IHPSYTNYR_vs_CRIS3_AVSPPAR
133 и 78
0,028
0,610
CFAB_YGLVTYATYPK_vs_PSG9_LFIPQITR
64 и 136
0,039
0,603
PSG2_IHPSYTNYR_vs_CRIS3_YEDLYSNCK
133 и 79
0,021
0,616
CFAB_YGLVTYATYPK_vs_SHBG_IALGGLLFPASNLR
64 и 18
0,045
0,601
PSG2_IHPSYTNYR_vs_FBLN1_TGYYFDGISR
133 и 86
0,024
0,613
CFAB_YGLVTYATYPK_vs_TENX_LNWEAPPGAFDSFLLR
64 и 141
0,019
0,617
PSG2_IHPSYTNYR_vs_LYAM1_SYYWIGIR
133 и 120
0,026
0,612
CFAB_YGLVTYATYPK_vs_TENX_LSQLSVTDVTTSSLR
64 и 142
0,036
0,605
PSG2_IHPSYTNYR_vs_PSG3_VSAPSGTGHLPGLNPL
133 и 134
0,011
0,628
CFAB_YGLVTYATYPK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
64 и 144
0,008
0,632
PSG2_IHPSYTNYR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
133 и 135
0,003
0,647
CLUS_ASSIIDELFQDR_vs_CRIS3_YEDLYSNCK
67 и 79
0,026
0,612
PSG2_IHPSYTNYR_vs_PSG9_LFIPQITR
133 и 136
0,012
0,626
CLUS_ASSIIDELFQDR_vs_LYAM1_SYYWIGIR
67 и 120
0,028
0,610
PSG2_IHPSYTNYR_vs_SHBG_IALGGLLFPASNLR
133 и 18
0,044
0,601
CLUS_ASSIIDELFQDR_vs_PSG3_VSAPSGTGHLPGLNPL
67 и 134
0,023
0,614
PSG2_IHPSYTNYR_vs_TENX_LNWEAPPGAFDSFLLR
133 и 141
0,028
0,610
CLUS_ASSIIDELFQDR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
67 и 135
0,012
0,626
PSG2_IHPSYTNYR_vs_TENX_LSQLSVTDVTTSSLR
133 и 142
0,020
0,617
CLUS_ASSIIDELFQDR_vs_TENX_LNWEAPPGAFDSFLLR
67 и 141
0,028
0,610
PTGDS_GPGEDFR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
137 и 135
0,026
0,611
CLUS_ASSIIDELFQDR_vs_TENX_LSQLSVTDVTTSSLR
67 и 142
0,036
0,605
PTGDS_GPGEDFR_vs_TENX_LNWEAPPGAFDSFLLR
137 и 141
0,034
0,607
CLUS_LFDSDPITVTVPVEVSR_vs_CRIS3_YEDLYSNCK
68 и 79
0,035
0,606
THBG_AVLHIGEK_vs_LYAM1_SYYWIGIR
143 и 120
0,034
0,606
CLUS_LFDSDPITVTVPVEVSR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
68 и 135
0,019
0,617
THBG_AVLHIGEK_vs_PSG3_VSAPSGTGHLPGLNPL
143 и 134
0,045
0,601
CO5_TLLPVSKPEIR_vs_CRIS3_AVSPPAR
70 и 78
0,023
0,614
THBG_AVLHIGEK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
143 и 135
0,020
0,617
CO5_TLLPVSKPEIR_vs_CRIS3_YEDLYSNCK
70 и 79
0,015
0,622
VTNC_GQYCYELDEK_vs_C163A_INPASLDK
149 и 54
0,024
0,613
CO5_TLLPVSKPEIR_vs_LYAM1_SYYWIGIR
70 и 120
0,014
0,623
VTNC_GQYCYELDEK_vs_CHL1_VIAVNEVGR
149 и 66
0,007
0,635
CO5_TLLPVSKPEIR_vs_PSG3_VSAPSGTGHLPGLNPL
70 и 134
0,003
0,650
VTNC_GQYCYELDEK_vs_CRIS3_AVSPPAR
149 и 78
0,006
0,638
CO5_TLLPVSKPEIR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
70 и 135
0,022
0,615
VTNC_GQYCYELDEK_vs_CRIS3_YEDLYSNCK
149 и 79
0,004
0,645
CO5_TLLPVSKPEIR_vs_SHBG_IALGGLLFPASNLR
70 и 18
0,025
0,613
VTNC_GQYCYELDEK_vs_FBLN1_TGYYFDGISR
149 и 86
0,040
0,603
CO5_TLLPVSKPEIR_vs_SPRL1_VLTHSELAPLR
70 и 140
0,041
0,602
VTNC_GQYCYELDEK_vs_IBP3_FLNVLSPR
149 и 99
0,032
0,608
CO5_TLLPVSKPEIR_vs_TENX_LNWEAPPGAFDSFLLR
70 и 141
0,014
0,623
VTNC_GQYCYELDEK_vs_IBP3_YGQPLPGYTTK
149 и 100
0,013
0,624
CO5_TLLPVSKPEIR_vs_TENX_LSQLSVTDVTTSSLR
70 и 142
0,020
0,617
VTNC_GQYCYELDEK_vs_IGF2_GIVEECCFR
149 и 103
0,011
0,627
CO5_VFQFLEK_vs_CRIS3_AVSPPAR
71 и 78
0,029
0,610
VTNC_GQYCYELDEK_vs_ITIH4_ILDDLSPR
149 и 112
0,034
0,606
CO5_VFQFLEK_vs_CRIS3_YEDLYSNCK
71 и 79
0,019
0,618
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,001
0,664
CO5_VFQFLEK_vs_LYAM1_SYYWIGIR
71 и 120
0,017
0,620
VTNC_GQYCYELDEK_vs_NCAM1_GLGEISAASEFK
149 и 121
0,014
0,623
CO5_VFQFLEK_vs_PSG3_VSAPSGTGHLPGLNPL
71 и 134
0,009
0,630
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,007
0,635
CO5_VFQFLEK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
71 и 135
0,021
0,615
VTNC_GQYCYELDEK_vs_PSG3_VSAPSGTGHLPGLNPL
149 и 134
0,000
0,681
CO5_VFQFLEK_vs_SHBG_IALGGLLFPASNLR
71 и 18
0,030
0,609
VTNC_GQYCYELDEK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
149 и 135
0,005
0,639
CO5_VFQFLEK_vs_TENX_LNWEAPPGAFDSFLLR
71 и 141
0,013
0,624
VTNC_GQYCYELDEK_vs_PSG9_LFIPQITR
149 и 136
0,016
0,621
CO5_VFQFLEK_vs_TENX_LSQLSVTDVTTSSLR
71 и 142
0,028
0,610
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,002
0,654
CO5_VFQFLEK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
71 и 144
0,035
0,606
VTNC_GQYCYELDEK_vs_SPRL1_VLTHSELAPLR
149 и 140
0,006
0,637
CO6_ALNHLPLEYNSALYSR_vs_CHL1_VIAVNEVGR
72 и 66
0,045
0,601
VTNC_GQYCYELDEK_vs_TENX_LNWEAPPGAFDSFLLR
149 и 141
0,004
0,644
CO6_ALNHLPLEYNSALYSR_vs_PSG3_VSAPSGTGHLPGLNPL
72 и 134
0,026
0,611
VTNC_GQYCYELDEK_vs_TENX_LSQLSVTDVTTSSLR
149 и 142
0,005
0,639
CO6_ALNHLPLEYNSALYSR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
72 и 135
0,028
0,611
VTNC_GQYCYELDEK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
149 и 144
0,003
0,650
CO6_ALNHLPLEYNSALYSR_vs_TENX_LNWEAPPGAFDSFLLR
72 и 141
0,024
0,613
VTNC_GQYCYELDEK_vs_VTDB_ELPEHTVK
149 и 147
0,003
0,647
CO6_ALNHLPLEYNSALYSR_vs_TENX_LSQLSVTDVTTSSLR
72 и 142
0,034
0,606
VTNC_VDTVDPPYPR_vs_C163A_INPASLDK
150 и 54
0,037
0,604
CO8A_SLLQPNK_vs_CHL1_VIAVNEVGR
74 и 66
0,016
0,621
VTNC_VDTVDPPYPR_vs_CHL1_VIAVNEVGR
150 и 66
0,013
0,625
CO8A_SLLQPNK_vs_CRIS3_AVSPPAR
74 и 78
0,035
0,606
VTNC_VDTVDPPYPR_vs_CRIS3_AVSPPAR
150 и 78
0,006
0,637
CO8A_SLLQPNK_vs_CRIS3_YEDLYSNCK
74 и 79
0,014
0,623
VTNC_VDTVDPPYPR_vs_CRIS3_YEDLYSNCK
150 и 79
0,004
0,645
CO8A_SLLQPNK_vs_LYAM1_SYYWIGIR
74 и 120
0,019
0,618
VTNC_VDTVDPPYPR_vs_IGF2_GIVEECCFR
150 и 103
0,032
0,608
CO8A_SLLQPNK_vs_PSG3_VSAPSGTGHLPGLNPL
74 и 134
0,003
0,649
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,001
0,663
CO8A_SLLQPNK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
74 и 135
0,017
0,620
VTNC_VDTVDPPYPR_vs_NCAM1_GLGEISAASEFK
150 и 121
0,015
0,622
CO8A_SLLQPNK_vs_PSG9_LFIPQITR
74 и 136
0,038
0,604
VTNC_VDTVDPPYPR_vs_PGRP2_AGLLRPDYALLGHR
150 и 126
0,018
0,619
CO8A_SLLQPNK_vs_SHBG_IALGGLLFPASNLR
74 и 18
0,032
0,607
VTNC_VDTVDPPYPR_vs_PSG3_VSAPSGTGHLPGLNPL
150 и 134
0,000
0,676
CO8A_SLLQPNK_vs_SPRL1_VLTHSELAPLR
74 и 140
0,013
0,624
VTNC_VDTVDPPYPR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
150 и 135
0,006
0,637
CO8A_SLLQPNK_vs_TENX_LNWEAPPGAFDSFLLR
74 и 141
0,004
0,646
VTNC_VDTVDPPYPR_vs_PSG9_LFIPQITR
150 и 136
0,020
0,617
CO8A_SLLQPNK_vs_TENX_LSQLSVTDVTTSSLR
74 и 142
0,007
0,635
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,004
0,643
CO8A_SLLQPNK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
74 и 144
0,012
0,626
VTNC_VDTVDPPYPR_vs_SPRL1_VLTHSELAPLR
150 и 140
0,009
0,630
CO8B_QALEEFQK_vs_CHL1_VIAVNEVGR
76 и 66
0,016
0,621
VTNC_VDTVDPPYPR_vs_TENX_LNWEAPPGAFDSFLLR
150 и 141
0,009
0,631
CO8B_QALEEFQK_vs_CRIS3_AVSPPAR
76 и 78
0,040
0,603
VTNC_VDTVDPPYPR_vs_TENX_LSQLSVTDVTTSSLR
150 и 142
0,010
0,629
CO8B_QALEEFQK_vs_CRIS3_YEDLYSNCK
76 и 79
0,015
0,622
VTNC_VDTVDPPYPR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
150 и 144
0,008
0,634
CO8B_QALEEFQK_vs_IGF2_GIVEECCFR
76 и 103
0,034
0,606
VTNC_VDTVDPPYPR_vs_VTDB_ELPEHTVK
150 и 147
0,002
0,652
CO8B_QALEEFQK_vs_LYAM1_SYYWIGIR
76 и 120
0,035
0,606
Таблица 53: эффективность классификации по обращениям, недели 18, 19 и 20.
AUROC обращения для недель беременности от 18 0/7 до 20 6/7 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, без BMI-стратификации.
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,037
0,636
CLUS_LFDSDPITVTVPVEVSR_vs_CRIS3_YEDLYSNCK
68 и 79
0,008
0,674
AFAM_DADPDTFFAK_vs_LYAM1_SYYWIGIR
37 и 120
0,021
0,651
CLUS_LFDSDPITVTVPVEVSR_vs_CSH_AHQLAIDTYQEFEETYIPK
68 и 80
0,029
0,643
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,034
0,639
CLUS_LFDSDPITVTVPVEVSR_vs_FBLN1_TGYYFDGISR
68 и 86
0,041
0,634
AFAM_HFQNLGK_vs_LYAM1_SYYWIGIR
38 и 120
0,026
0,645
CLUS_LFDSDPITVTVPVEVSR_vs_IBP3_FLNVLSPR
68 и 99
0,043
0,632
ANGT_DPTFIPAPIQAK_vs_LYAM1_SYYWIGIR
42 и 120
0,018
0,654
CLUS_LFDSDPITVTVPVEVSR_vs_IBP3_YGQPLPGYTTK
68 и 100
0,028
0,644
APOC3_GWVTDGFSSLK_vs_ALS_IRPHTFTGLSGLR
47 и 40
0,030
0,642
CLUS_LFDSDPITVTVPVEVSR_vs_IGF2_GIVEECCFR
68 и 103
0,007
0,675
APOC3_GWVTDGFSSLK_vs_C163A_INPASLDK
47 и 54
0,016
0,658
CLUS_LFDSDPITVTVPVEVSR_vs_LYAM1_SYYWIGIR
68 и 120
0,001
0,717
APOC3_GWVTDGFSSLK_vs_CRIS3_AVSPPAR
47 и 78
0,013
0,662
CLUS_LFDSDPITVTVPVEVSR_vs_PSG3_VSAPSGTGHLPGLNPL
68 и 134
0,019
0,653
APOC3_GWVTDGFSSLK_vs_CRIS3_YEDLYSNCK
47 и 79
0,010
0,669
CLUS_LFDSDPITVTVPVEVSR_vs_SHBG_IALGGLLFPASNLR
68 и 18
0,015
0,658
APOC3_GWVTDGFSSLK_vs_CSH_AHQLAIDTYQEFEETYIPK
47 и 80
0,020
0,652
CLUS_LFDSDPITVTVPVEVSR_vs_SPRL1_VLTHSELAPLR
68 и 140
0,048
0,629
APOC3_GWVTDGFSSLK_vs_CSH_ISLLLIESWLEPVR
47 и 81
0,037
0,636
CLUS_LFDSDPITVTVPVEVSR_vs_TENX_LNWEAPPGAFDSFLLR
68 и 141
0,026
0,646
APOC3_GWVTDGFSSLK_vs_FBLN1_TGYYFDGISR
47 и 86
0,034
0,639
CLUS_LFDSDPITVTVPVEVSR_vs_TENX_LSQLSVTDVTTSSLR
68 и 142
0,044
0,632
APOC3_GWVTDGFSSLK_vs_IBP3_FLNVLSPR
47 и 99
0,023
0,649
CLUS_LFDSDPITVTVPVEVSR_vs_VTDB_ELPEHTVK
68 и 147
0,002
0,705
APOC3_GWVTDGFSSLK_vs_IBP3_YGQPLPGYTTK
47 и 100
0,024
0,648
CO5_TLLPVSKPEIR_vs_CRIS3_AVSPPAR
70 и 78
0,038
0,635
APOC3_GWVTDGFSSLK_vs_IGF2_GIVEECCFR
47 и 103
0,025
0,646
CO5_TLLPVSKPEIR_vs_CRIS3_YEDLYSNCK
70 и 79
0,027
0,644
APOC3_GWVTDGFSSLK_vs_ITIH4_ILDDLSPR
47 и 112
0,037
0,636
CO5_TLLPVSKPEIR_vs_LYAM1_SYYWIGIR
70 и 120
0,005
0,683
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,003
0,692
CO5_VFQFLEK_vs_CRIS3_YEDLYSNCK
71 и 79
0,047
0,630
APOC3_GWVTDGFSSLK_vs_PGRP2_AGLLRPDYALLGHR
47 и 126
0,034
0,639
CO5_VFQFLEK_vs_LYAM1_SYYWIGIR
71 и 120
0,008
0,674
APOC3_GWVTDGFSSLK_vs_PSG3_VSAPSGTGHLPGLNPL
47 и 134
0,042
0,633
CO6_ALNHLPLEYNSALYSR_vs_CRIS3_YEDLYSNCK
72 и 79
0,042
0,633
APOC3_GWVTDGFSSLK_vs_SHBG_IALGGLLFPASNLR
47 и 18
0,025
0,647
CO6_ALNHLPLEYNSALYSR_vs_LYAM1_SYYWIGIR
72 и 120
0,005
0,684
APOC3_GWVTDGFSSLK_vs_SOM2.CSH_NYGLLYCFR
47 и 138
0,034
0,638
CO6_ALNHLPLEYNSALYSR_vs_TENX_LNWEAPPGAFDSFLLR
72 и 141
0,048
0,629
APOC3_GWVTDGFSSLK_vs_SOM2.CSH_SVEGSCGF
47 и 139
0,025
0,647
CO6_ALNHLPLEYNSALYSR_vs_VTDB_ELPEHTVK
72 и 147
0,042
0,633
APOC3_GWVTDGFSSLK_vs_SPRL1_VLTHSELAPLR
47 и 140
0,033
0,640
CO8A_SLLQPNK_vs_CRIS3_YEDLYSNCK
74 и 79
0,032
0,640
APOC3_GWVTDGFSSLK_vs_TENX_LNWEAPPGAFDSFLLR
47 и 141
0,043
0,632
CO8A_SLLQPNK_vs_IGF2_GIVEECCFR
74 и 103
0,046
0,630
APOC3_GWVTDGFSSLK_vs_TENX_LSQLSVTDVTTSSLR
47 и 142
0,038
0,636
CO8A_SLLQPNK_vs_LYAM1_SYYWIGIR
74 и 120
0,003
0,693
APOC3_GWVTDGFSSLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
47 и 144
0,023
0,648
CO8A_SLLQPNK_vs_PSG3_VSAPSGTGHLPGLNPL
74 и 134
0,019
0,653
APOC3_GWVTDGFSSLK_vs_VTDB_ELPEHTVK
47 и 147
0,024
0,648
CO8A_SLLQPNK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
74 и 144
0,028
0,644
APOH_ATVVYQGER_vs_CRIS3_AVSPPAR
48 и 78
0,025
0,646
CO8A_SLLQPNK_vs_VTDB_ELPEHTVK
74 и 147
0,047
0,630
APOH_ATVVYQGER_vs_CRIS3_YEDLYSNCK
48 и 79
0,014
0,660
CO8B_QALEEFQK_vs_CRIS3_YEDLYSNCK
76 и 79
0,031
0,641
APOH_ATVVYQGER_vs_IGF2_GIVEECCFR
48 и 103
0,040
0,634
CO8B_QALEEFQK_vs_IGF2_GIVEECCFR
76 и 103
0,044
0,631
APOH_ATVVYQGER_vs_LYAM1_SYYWIGIR
48 и 120
0,003
0,691
CO8B_QALEEFQK_vs_LYAM1_SYYWIGIR
76 и 120
0,012
0,664
APOH_ATVVYQGER_vs_PSG3_VSAPSGTGHLPGLNPL
48 и 134
0,030
0,642
CO8B_QALEEFQK_vs_PSG3_VSAPSGTGHLPGLNPL
76 и 134
0,023
0,649
APOH_ATVVYQGER_vs_SHBG_IALGGLLFPASNLR
48 и 18
0,045
0,631
CO8B_QALEEFQK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
76 и 144
0,033
0,640
APOH_ATVVYQGER_vs_VTDB_ELPEHTVK
48 и 147
0,011
0,666
F13B_GDTYPAELYITGSILR_vs_CRIS3_AVSPPAR
84 и 78
0,022
0,649
B2MG_VEHSDLSFSK_vs_LYAM1_SYYWIGIR
50 и 120
0,025
0,646
F13B_GDTYPAELYITGSILR_vs_CRIS3_YEDLYSNCK
84 и 79
0,014
0,661
B2MG_VNHVTLSQPK_vs_C163A_INPASLDK
51 и 54
0,033
0,639
F13B_GDTYPAELYITGSILR_vs_FBLN1_TGYYFDGISR
84 и 86
0,048
0,629
B2MG_VNHVTLSQPK_vs_CRIS3_AVSPPAR
51 и 78
0,039
0,635
F13B_GDTYPAELYITGSILR_vs_IGF2_GIVEECCFR
84 и 103
0,040
0,634
B2MG_VNHVTLSQPK_vs_CRIS3_YEDLYSNCK
51 и 79
0,024
0,647
F13B_GDTYPAELYITGSILR_vs_LYAM1_SYYWIGIR
84 и 120
0,001
0,711
B2MG_VNHVTLSQPK_vs_LYAM1_SYYWIGIR
51 и 120
0,003
0,692
F13B_GDTYPAELYITGSILR_vs_PSG3_VSAPSGTGHLPGLNPL
84 и 134
0,034
0,638
BGH3_LTLLAPLNSVFK_vs_LYAM1_SYYWIGIR
52 и 120
0,018
0,655
F13B_GDTYPAELYITGSILR_vs_SHBG_IALGGLLFPASNLR
84 и 18
0,045
0,631
CATD_VGFAEAAR_vs_C163A_INPASLDK
57 и 54
0,008
0,674
FETUA_FSVVYAK_vs_LYAM1_SYYWIGIR
88 и 120
0,041
0,633
CATD_VGFAEAAR_vs_CRIS3_AVSPPAR
57 и 78
0,046
0,631
HABP2_FLNWIK_vs_C163A_INPASLDK
92 и 54
0,025
0,647
CATD_VGFAEAAR_vs_CRIS3_YEDLYSNCK
57 и 79
0,030
0,642
HABP2_FLNWIK_vs_CRIS3_AVSPPAR
92 и 78
0,036
0,637
CATD_VGFAEAAR_vs_CSH_AHQLAIDTYQEFEETYIPK
57 и 80
0,042
0,633
HABP2_FLNWIK_vs_CRIS3_YEDLYSNCK
92 и 79
0,023
0,649
CATD_VGFAEAAR_vs_LYAM1_SYYWIGIR
57 и 120
0,009
0,670
HABP2_FLNWIK_vs_CSH_AHQLAIDTYQEFEETYIPK
92 и 80
0,033
0,639
CATD_VGFAEAAR_vs_SOM2.CSH_SVEGSCGF
57 и 139
0,047
0,630
HABP2_FLNWIK_vs_FBLN1_TGYYFDGISR
92 и 86
0,021
0,651
CATD_VGFAEAAR_vs_TENX_LNWEAPPGAFDSFLLR
57 и 141
0,028
0,643
HABP2_FLNWIK_vs_IGF2_GIVEECCFR
92 и 103
0,018
0,655
CATD_VGFAEAAR_vs_TENX_LSQLSVTDVTTSSLR
57 и 142
0,046
0,630
HABP2_FLNWIK_vs_ITIH4_ILDDLSPR
92 и 112
0,028
0,644
CATD_VSTLPAITLK_vs_C163A_INPASLDK
58 и 54
0,035
0,638
HABP2_FLNWIK_vs_LYAM1_SYYWIGIR
92 и 120
0,002
0,699
CATD_VSTLPAITLK_vs_CRIS3_AVSPPAR
58 и 78
0,047
0,630
HABP2_FLNWIK_vs_PGRP2_AGLLRPDYALLGHR
92 и 126
0,023
0,649
CATD_VSTLPAITLK_vs_CRIS3_YEDLYSNCK
58 и 79
0,032
0,640
HABP2_FLNWIK_vs_PSG3_VSAPSGTGHLPGLNPL
92 и 134
0,009
0,670
CATD_VSTLPAITLK_vs_LYAM1_SYYWIGIR
58 и 120
0,013
0,663
HABP2_FLNWIK_vs_SHBG_IALGGLLFPASNLR
92 и 18
0,015
0,659
CATD_VSTLPAITLK_vs_PSG3_VSAPSGTGHLPGLNPL
58 и 134
0,046
0,630
HABP2_FLNWIK_vs_SOM2.CSH_NYGLLYCFR
92 и 138
0,037
0,637
CATD_VSTLPAITLK_vs_TENX_LNWEAPPGAFDSFLLR
58 и 141
0,035
0,638
HABP2_FLNWIK_vs_SOM2.CSH_SVEGSCGF
92 и 139
0,025
0,647
CBPN_EALIQFLEQVHQGIK_vs_LYAM1_SYYWIGIR
59 и 120
0,047
0,630
HABP2_FLNWIK_vs_TENX_LNWEAPPGAFDSFLLR
92 и 141
0,017
0,656
CBPN_NNANGVDLNR_vs_LYAM1_SYYWIGIR
60 и 120
0,013
0,662
HABP2_FLNWIK_vs_TENX_LSQLSVTDVTTSSLR
92 и 142
0,021
0,651
CBPN_NNANGVDLNR_vs_SPRL1_VLTHSELAPLR
60 и 140
0,043
0,632
HABP2_FLNWIK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
92 и 144
0,011
0,665
CD14_LTVGAAQVPAQLLVGALR_vs_C163A_INPASLDK
61 и 54
0,041
0,633
HABP2_FLNWIK_vs_VTDB_ELPEHTVK
92 и 147
0,002
0,704
CD14_LTVGAAQVPAQLLVGALR_vs_CRIS3_AVSPPAR
61 и 78
0,044
0,632
HEMO_NFPSPVDAAFR_vs_LYAM1_SYYWIGIR
93 и 120
0,019
0,653
CD14_LTVGAAQVPAQLLVGALR_vs_CRIS3_YEDLYSNCK
61 и 79
0,021
0,651
IBP4_QCHPALDGQR_vs_CRIS3_AVSPPAR
2 и 78
0,012
0,665
CD14_LTVGAAQVPAQLLVGALR_vs_CSH_AHQLAIDTYQEFEETYIPK
61 и 80
0,013
0,662
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,008
0,674
CD14_LTVGAAQVPAQLLVGALR_vs_CSH_ISLLLIESWLEPVR
61 и 81
0,038
0,636
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,002
0,705
CD14_LTVGAAQVPAQLLVGALR_vs_FBLN1_TGYYFDGISR
61 и 86
0,024
0,647
IBP4_QCHPALDGQR_vs_PSG3_VSAPSGTGHLPGLNPL
2 и 134
0,020
0,652
CD14_LTVGAAQVPAQLLVGALR_vs_IBP1_VVESLAK
61 и 97
0,043
0,633
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,039
0,635
CD14_LTVGAAQVPAQLLVGALR_vs_IGF2_GIVEECCFR
61 и 103
0,022
0,650
IBP4_QCHPALDGQR_vs_SPRL1_VLTHSELAPLR
2 и 140
0,048
0,629
CD14_LTVGAAQVPAQLLVGALR_vs_ITIH4_ILDDLSPR
61 и 112
0,037
0,637
IBP6_HLDSVLQQLQTEVYR_vs_LYAM1_SYYWIGIR
102 и 120
0,031
0,641
CD14_LTVGAAQVPAQLLVGALR_vs_LYAM1_SYYWIGIR
61 и 120
0,001
0,717
KNG1_DIPTNSPELEETLTHTITK_vs_CRIS3_YEDLYSNCK
116 и 79
0,042
0,633
CD14_LTVGAAQVPAQLLVGALR_vs_PGRP2_AGLLRPDYALLGHR
61 и 126
0,017
0,655
KNG1_DIPTNSPELEETLTHTITK_vs_LYAM1_SYYWIGIR
116 и 120
0,007
0,678
CD14_LTVGAAQVPAQLLVGALR_vs_PSG3_VSAPSGTGHLPGLNPL
61 и 134
0,008
0,673
KNG1_QVVAGLNFR_vs_CRIS3_AVSPPAR
117 и 78
0,040
0,634
CD14_LTVGAAQVPAQLLVGALR_vs_SHBG_IALGGLLFPASNLR
61 и 18
0,015
0,659
KNG1_QVVAGLNFR_vs_CRIS3_YEDLYSNCK
117 и 79
0,020
0,652
CD14_LTVGAAQVPAQLLVGALR_vs_SOM2.CSH_SVEGSCGF
61 и 139
0,020
0,652
KNG1_QVVAGLNFR_vs_CSH_AHQLAIDTYQEFEETYIPK
117 и 80
0,025
0,647
CD14_LTVGAAQVPAQLLVGALR_vs_SPRL1_VLTHSELAPLR
61 и 140
0,026
0,646
KNG1_QVVAGLNFR_vs_IGF2_GIVEECCFR
117 и 103
0,040
0,634
CD14_LTVGAAQVPAQLLVGALR_vs_TENX_LNWEAPPGAFDSFLLR
61 и 141
0,024
0,647
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,002
0,703
CD14_LTVGAAQVPAQLLVGALR_vs_TENX_LSQLSVTDVTTSSLR
61 и 142
0,020
0,652
KNG1_QVVAGLNFR_vs_PSG3_VSAPSGTGHLPGLNPL
117 и 134
0,039
0,635
CD14_LTVGAAQVPAQLLVGALR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
61 и 144
0,008
0,673
KNG1_QVVAGLNFR_vs_SHBG_IALGGLLFPASNLR
117 и 18
0,029
0,642
CD14_LTVGAAQVPAQLLVGALR_vs_VTDB_ELPEHTVK
61 и 147
0,002
0,700
LBP_ITGFLKPGK_vs_LYAM1_SYYWIGIR
118 и 120
0,050
0,628
CD14_SWLAELQQWLKPGLK_vs_C163A_INPASLDK
62 и 54
0,041
0,634
LBP_ITLPDFTGDLR_vs_LYAM1_SYYWIGIR
119 и 120
0,048
0,629
CD14_SWLAELQQWLKPGLK_vs_CRIS3_AVSPPAR
62 и 78
0,047
0,630
PEDF_LQSLFDSPDFSK_vs_CRIS3_AVSPPAR
124 и 78
0,031
0,641
CD14_SWLAELQQWLKPGLK_vs_CRIS3_YEDLYSNCK
62 и 79
0,028
0,643
PEDF_LQSLFDSPDFSK_vs_CRIS3_YEDLYSNCK
124 и 79
0,024
0,647
CD14_SWLAELQQWLKPGLK_vs_CSH_AHQLAIDTYQEFEETYIPK
62 и 80
0,027
0,645
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,007
0,677
CD14_SWLAELQQWLKPGLK_vs_FBLN1_TGYYFDGISR
62 и 86
0,035
0,638
PEDF_TVQAVLTVPK_vs_CRIS3_AVSPPAR
125 и 78
0,023
0,648
CD14_SWLAELQQWLKPGLK_vs_IGF2_GIVEECCFR
62 и 103
0,039
0,635
PEDF_TVQAVLTVPK_vs_CRIS3_YEDLYSNCK
125 и 79
0,017
0,656
CD14_SWLAELQQWLKPGLK_vs_ITIH4_ILDDLSPR
62 и 112
0,036
0,637
PEDF_TVQAVLTVPK_vs_LYAM1_SYYWIGIR
125 и 120
0,003
0,692
CD14_SWLAELQQWLKPGLK_vs_LYAM1_SYYWIGIR
62 и 120
0,002
0,701
PSG2_IHPSYTNYR_vs_CRIS3_AVSPPAR
133 и 78
0,015
0,659
CD14_SWLAELQQWLKPGLK_vs_PGRP2_AGLLRPDYALLGHR
62 и 126
0,027
0,644
PSG2_IHPSYTNYR_vs_CRIS3_YEDLYSNCK
133 и 79
0,011
0,666
CD14_SWLAELQQWLKPGLK_vs_PSG3_VSAPSGTGHLPGLNPL
62 и 134
0,014
0,660
PSG2_IHPSYTNYR_vs_CSH_AHQLAIDTYQEFEETYIPK
133 и 80
0,036
0,637
CD14_SWLAELQQWLKPGLK_vs_SHBG_IALGGLLFPASNLR
62 и 18
0,021
0,650
PSG2_IHPSYTNYR_vs_FBLN1_TGYYFDGISR
133 и 86
0,006
0,680
CD14_SWLAELQQWLKPGLK_vs_SOM2.CSH_SVEGSCGF
62 и 139
0,024
0,647
PSG2_IHPSYTNYR_vs_IBP1_VVESLAK
133 и 97
0,050
0,628
CD14_SWLAELQQWLKPGLK_vs_SPRL1_VLTHSELAPLR
62 и 140
0,036
0,637
PSG2_IHPSYTNYR_vs_IGF2_GIVEECCFR
133 и 103
0,047
0,630
CD14_SWLAELQQWLKPGLK_vs_TENX_LNWEAPPGAFDSFLLR
62 и 141
0,041
0,634
PSG2_IHPSYTNYR_vs_LYAM1_SYYWIGIR
133 и 120
0,008
0,674
CD14_SWLAELQQWLKPGLK_vs_TENX_LSQLSVTDVTTSSLR
62 и 142
0,033
0,639
PSG2_IHPSYTNYR_vs_PSG3_VSAPSGTGHLPGLNPL
133 и 134
0,011
0,665
CD14_SWLAELQQWLKPGLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
62 и 144
0,013
0,662
PSG2_IHPSYTNYR_vs_SHBG_IALGGLLFPASNLR
133 и 18
0,021
0,651
CD14_SWLAELQQWLKPGLK_vs_VTDB_ELPEHTVK
62 и 147
0,004
0,686
PSG2_IHPSYTNYR_vs_SPRL1_VLTHSELAPLR
133 и 140
0,037
0,637
CFAB_YGLVTYATYPK_vs_LYAM1_SYYWIGIR
64 и 120
0,049
0,629
PTGDS_GPGEDFR_vs_CRIS3_AVSPPAR
137 и 78
0,039
0,635
CLUS_ASSIIDELFQDR_vs_CRIS3_AVSPPAR
67 и 78
0,026
0,646
PTGDS_GPGEDFR_vs_CRIS3_YEDLYSNCK
137 и 79
0,029
0,642
CLUS_ASSIIDELFQDR_vs_CRIS3_YEDLYSNCK
67 и 79
0,018
0,654
PTGDS_GPGEDFR_vs_LYAM1_SYYWIGIR
137 и 120
0,013
0,662
CLUS_ASSIIDELFQDR_vs_CSH_AHQLAIDTYQEFEETYIPK
67 и 80
0,042
0,633
THBG_AVLHIGEK_vs_LYAM1_SYYWIGIR
143 и 120
0,011
0,665
CLUS_ASSIIDELFQDR_vs_FBLN1_TGYYFDGISR
67 и 86
0,029
0,643
VTNC_GQYCYELDEK_vs_CRIS3_YEDLYSNCK
149 и 79
0,039
0,635
CLUS_ASSIIDELFQDR_vs_IGF2_GIVEECCFR
67 и 103
0,013
0,663
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,003
0,697
CLUS_ASSIIDELFQDR_vs_LYAM1_SYYWIGIR
67 и 120
0,001
0,712
VTNC_GQYCYELDEK_vs_PSG3_VSAPSGTGHLPGLNPL
149 и 134
0,021
0,651
CLUS_ASSIIDELFQDR_vs_PSG3_VSAPSGTGHLPGLNPL
67 и 134
0,015
0,660
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,047
0,630
CLUS_ASSIIDELFQDR_vs_SHBG_IALGGLLFPASNLR
67 и 18
0,022
0,650
VTNC_GQYCYELDEK_vs_VTDB_ELPEHTVK
149 и 147
0,045
0,631
CLUS_ASSIIDELFQDR_vs_TENX_LNWEAPPGAFDSFLLR
67 и 141
0,048
0,629
VTNC_VDTVDPPYPR_vs_CRIS3_AVSPPAR
150 и 78
0,042
0,633
CLUS_ASSIIDELFQDR_vs_VTDB_ELPEHTVK
67 и 147
0,005
0,682
VTNC_VDTVDPPYPR_vs_CRIS3_YEDLYSNCK
150 и 79
0,035
0,638
CLUS_LFDSDPITVTVPVEVSR_vs_C163A_INPASLDK
68 и 54
0,046
0,631
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,004
0,687
CLUS_LFDSDPITVTVPVEVSR_vs_CRIS3_AVSPPAR
68 и 78
0,013
0,662
VTNC_VDTVDPPYPR_vs_VTDB_ELPEHTVK
150 и 147
0,036
0,637
Таблица 54: эффективность классификации по обращениям, недели 18, 19 и 20.
AUROC обращения для недель беременности от 18 0/7 до 20 6/7 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, с BMI-стратификацией (>22≤37).
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
A2GL_DLLLPQPDLR_vs_LYAM1_SYYWIGIR
34 и 120
0,017
0,699
F13B_GDTYPAELYITGSILR_vs_LYAM1_SYYWIGIR
84 и 120
0,001
0,768
AFAM_DADPDTFFAK_vs_CRIS3_YEDLYSNCK
37 и 79
0,031
0,679
HABP2_FLNWIK_vs_LYAM1_SYYWIGIR
92 и 120
0,012
0,710
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,036
0,674
HEMO_NFPSPVDAAFR_vs_LYAM1_SYYWIGIR
93 и 120
0,027
0,683
AFAM_DADPDTFFAK_vs_LYAM1_SYYWIGIR
37 и 120
0,012
0,708
IBP4_QCHPALDGQR_vs_CRIS3_AVSPPAR
2 и 78
0,015
0,702
AFAM_DADPDTFFAK_vs_PGRP2_AGLLRPDYALLGHR
37 и 126
0,039
0,672
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,011
0,710
AFAM_DADPDTFFAK_vs_SOM2.CSH_SVEGSCGF
37 и 139
0,044
0,668
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,001
0,774
AFAM_HFQNLGK_vs_LYAM1_SYYWIGIR
38 и 120
0,018
0,697
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,045
0,666
AFAM_HFQNLGK_vs_SOM2.CSH_SVEGSCGF
38 и 139
0,046
0,666
IBP6_HLDSVLQQLQTEVYR_vs_LYAM1_SYYWIGIR
102 и 120
0,017
0,698
ANGT_DPTFIPAPIQAK_vs_CRIS3_YEDLYSNCK
42 и 79
0,044
0,667
KNG1_DIPTNSPELEETLTHTITK_vs_CRIS3_AVSPPAR
116 и 78
0,047
0,665
ANGT_DPTFIPAPIQAK_vs_LYAM1_SYYWIGIR
42 и 120
0,007
0,722
KNG1_DIPTNSPELEETLTHTITK_vs_CRIS3_YEDLYSNCK
116 и 79
0,045
0,667
APOC3_GWVTDGFSSLK_vs_CRIS3_AVSPPAR
47 и 78
0,044
0,667
KNG1_DIPTNSPELEETLTHTITK_vs_LYAM1_SYYWIGIR
116 и 120
0,005
0,735
APOC3_GWVTDGFSSLK_vs_CRIS3_YEDLYSNCK
47 и 79
0,043
0,668
KNG1_QVVAGLNFR_vs_CRIS3_AVSPPAR
117 и 78
0,026
0,685
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,019
0,694
KNG1_QVVAGLNFR_vs_CRIS3_YEDLYSNCK
117 и 79
0,018
0,696
APOH_ATVVYQGER_vs_CRIS3_AVSPPAR
48 и 78
0,022
0,690
KNG1_QVVAGLNFR_vs_IGF2_GIVEECCFR
117 и 103
0,048
0,665
APOH_ATVVYQGER_vs_CRIS3_YEDLYSNCK
48 и 79
0,013
0,706
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,002
0,761
APOH_ATVVYQGER_vs_LYAM1_SYYWIGIR
48 и 120
0,005
0,735
PAPP1_DIPHWLNPTR_vs_LYAM1_SYYWIGIR
122 и 120
0,031
0,679
APOH_ATVVYQGER_vs_PGRP2_AGLLRPDYALLGHR
48 и 126
0,014
0,704
PAPP1_DIPHWLNPTR_vs_PRG2_WNFAYWAAHQPWSR
122 и 129
0,033
0,677
APOH_ATVVYQGER_vs_VTDB_ELPEHTVK
48 и 147
0,011
0,712
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,010
0,714
B2MG_VEHSDLSFSK_vs_LYAM1_SYYWIGIR
50 и 120
0,040
0,671
PEDF_TVQAVLTVPK_vs_CRIS3_AVSPPAR
125 и 78
0,033
0,677
B2MG_VNHVTLSQPK_vs_CRIS3_AVSPPAR
51 и 78
0,035
0,675
PEDF_TVQAVLTVPK_vs_CRIS3_YEDLYSNCK
125 и 79
0,029
0,681
B2MG_VNHVTLSQPK_vs_CRIS3_YEDLYSNCK
51 и 79
0,027
0,684
PEDF_TVQAVLTVPK_vs_LYAM1_SYYWIGIR
125 и 120
0,004
0,739
B2MG_VNHVTLSQPK_vs_LYAM1_SYYWIGIR
51 и 120
0,004
0,741
PSG2_IHPSYTNYR_vs_ALS_IRPHTFTGLSGLR
133 и 40
0,016
0,700
BGH3_LTLLAPLNSVFK_vs_LYAM1_SYYWIGIR
52 и 120
0,033
0,677
PSG2_IHPSYTNYR_vs_C163A_INPASLDK
133 и 54
0,020
0,693
CATD_VGFAEAAR_vs_C163A_INPASLDK
57 и 54
0,020
0,693
PSG2_IHPSYTNYR_vs_CHL1_VIAVNEVGR
133 и 66
0,014
0,704
CATD_VGFAEAAR_vs_LYAM1_SYYWIGIR
57 и 120
0,015
0,701
PSG2_IHPSYTNYR_vs_CRIS3_AVSPPAR
133 и 78
0,001
0,764
CATD_VSTLPAITLK_vs_C163A_INPASLDK
58 и 54
0,039
0,672
PSG2_IHPSYTNYR_vs_CRIS3_YEDLYSNCK
133 и 79
0,002
0,759
CATD_VSTLPAITLK_vs_CRIS3_AVSPPAR
58 и 78
0,048
0,664
PSG2_IHPSYTNYR_vs_CSH_AHQLAIDTYQEFEETYIPK
133 и 80
0,008
0,721
CATD_VSTLPAITLK_vs_CRIS3_YEDLYSNCK
58 и 79
0,030
0,681
PSG2_IHPSYTNYR_vs_CSH_ISLLLIESWLEPVR
133 и 81
0,015
0,703
CATD_VSTLPAITLK_vs_LYAM1_SYYWIGIR
58 и 120
0,011
0,711
PSG2_IHPSYTNYR_vs_FBLN1_TGYYFDGISR
133 и 86
0,007
0,725
CBPN_NNANGVDLNR_vs_LYAM1_SYYWIGIR
60 и 120
0,017
0,698
PSG2_IHPSYTNYR_vs_IBP2_LIQGAPTIR
133 и 98
0,013
0,705
CD14_LTVGAAQVPAQLLVGALR_vs_CRIS3_AVSPPAR
61 и 78
0,028
0,683
PSG2_IHPSYTNYR_vs_IBP3_FLNVLSPR
133 и 99
0,013
0,707
CD14_LTVGAAQVPAQLLVGALR_vs_CRIS3_YEDLYSNCK
61 и 79
0,016
0,699
PSG2_IHPSYTNYR_vs_IBP3_YGQPLPGYTTK
133 и 100
0,017
0,699
CD14_LTVGAAQVPAQLLVGALR_vs_LYAM1_SYYWIGIR
61 и 120
0,001
0,777
PSG2_IHPSYTNYR_vs_IGF2_GIVEECCFR
133 и 103
0,010
0,713
CD14_LTVGAAQVPAQLLVGALR_vs_PGRP2_AGLLRPDYALLGHR
61 и 126
0,013
0,707
PSG2_IHPSYTNYR_vs_ITIH4_ILDDLSPR
133 и 112
0,008
0,719
CD14_LTVGAAQVPAQLLVGALR_vs_SHBG_IALGGLLFPASNLR
61 и 18
0,043
0,668
PSG2_IHPSYTNYR_vs_LYAM1_SYYWIGIR
133 и 120
0,000
0,790
CD14_LTVGAAQVPAQLLVGALR_vs_SOM2.CSH_SVEGSCGF
61 и 139
0,019
0,695
PSG2_IHPSYTNYR_vs_NCAM1_GLGEISAASEFK
133 и 121
0,043
0,668
CD14_LTVGAAQVPAQLLVGALR_vs_VTDB_ELPEHTVK
61 и 147
0,009
0,718
PSG2_IHPSYTNYR_vs_PGRP2_AGLLRPDYALLGHR
133 и 126
0,011
0,712
CD14_SWLAELQQWLKPGLK_vs_CRIS3_AVSPPAR
62 и 78
0,049
0,663
PSG2_IHPSYTNYR_vs_PSG3_VSAPSGTGHLPGLNPL
133 и 134
0,002
0,753
CD14_SWLAELQQWLKPGLK_vs_CRIS3_YEDLYSNCK
62 и 79
0,041
0,669
PSG2_IHPSYTNYR_vs_SHBG_IALGGLLFPASNLR
133 и 18
0,003
0,743
CD14_SWLAELQQWLKPGLK_vs_LYAM1_SYYWIGIR
62 и 120
0,005
0,732
PSG2_IHPSYTNYR_vs_SOM2.CSH_NYGLLYCFR
133 и 138
0,013
0,706
CD14_SWLAELQQWLKPGLK_vs_PGRP2_AGLLRPDYALLGHR
62 и 126
0,027
0,683
PSG2_IHPSYTNYR_vs_SOM2.CSH_SVEGSCGF
133 и 139
0,007
0,726
CD14_SWLAELQQWLKPGLK_vs_SOM2.CSH_SVEGSCGF
62 и 139
0,040
0,671
PSG2_IHPSYTNYR_vs_SPRL1_VLTHSELAPLR
133 и 140
0,018
0,697
CD14_SWLAELQQWLKPGLK_vs_VTDB_ELPEHTVK
62 и 147
0,040
0,671
PSG2_IHPSYTNYR_vs_TENX_LNWEAPPGAFDSFLLR
133 и 141
0,022
0,690
CLUS_ASSIIDELFQDR_vs_CRIS3_AVSPPAR
67 и 78
0,024
0,688
PSG2_IHPSYTNYR_vs_TENX_LSQLSVTDVTTSSLR
133 и 142
0,019
0,694
CLUS_ASSIIDELFQDR_vs_CRIS3_YEDLYSNCK
67 и 79
0,020
0,693
PSG2_IHPSYTNYR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
133 и 144
0,026
0,685
CLUS_ASSIIDELFQDR_vs_LYAM1_SYYWIGIR
67 и 120
0,004
0,736
PSG2_IHPSYTNYR_vs_VTDB_ELPEHTVK
133 и 147
0,005
0,732
CLUS_LFDSDPITVTVPVEVSR_vs_CRIS3_AVSPPAR
68 и 78
0,026
0,684
PTGDS_GPGEDFR_vs_C163A_INPASLDK
137 и 54
0,035
0,675
CLUS_LFDSDPITVTVPVEVSR_vs_CRIS3_YEDLYSNCK
68 и 79
0,019
0,694
PTGDS_GPGEDFR_vs_CRIS3_AVSPPAR
137 и 78
0,027
0,683
CLUS_LFDSDPITVTVPVEVSR_vs_LYAM1_SYYWIGIR
68 и 120
0,010
0,714
PTGDS_GPGEDFR_vs_CRIS3_YEDLYSNCK
137 и 79
0,022
0,691
CO5_TLLPVSKPEIR_vs_CRIS3_AVSPPAR
70 и 78
0,048
0,664
PTGDS_GPGEDFR_vs_LYAM1_SYYWIGIR
137 и 120
0,006
0,729
CO5_TLLPVSKPEIR_vs_CRIS3_YEDLYSNCK
70 и 79
0,039
0,671
PTGDS_GPGEDFR_vs_SOM2.CSH_SVEGSCGF
137 и 139
0,034
0,677
CO5_TLLPVSKPEIR_vs_LYAM1_SYYWIGIR
70 и 120
0,008
0,720
THBG_AVLHIGEK_vs_LYAM1_SYYWIGIR
143 и 120
0,019
0,695
CO5_VFQFLEK_vs_LYAM1_SYYWIGIR
71 и 120
0,015
0,701
VTNC_GQYCYELDEK_vs_CRIS3_AVSPPAR
149 и 78
0,031
0,679
CO6_ALNHLPLEYNSALYSR_vs_CRIS3_YEDLYSNCK
72 и 79
0,043
0,668
VTNC_GQYCYELDEK_vs_CRIS3_YEDLYSNCK
149 и 79
0,027
0,684
CO6_ALNHLPLEYNSALYSR_vs_LYAM1_SYYWIGIR
72 и 120
0,005
0,731
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,002
0,760
CO8A_SLLQPNK_vs_CRIS3_YEDLYSNCK
74 и 79
0,033
0,677
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,049
0,663
CO8A_SLLQPNK_vs_LYAM1_SYYWIGIR
74 и 120
0,005
0,735
VTNC_VDTVDPPYPR_vs_CRIS3_AVSPPAR
150 и 78
0,041
0,669
CO8B_QALEEFQK_vs_LYAM1_SYYWIGIR
76 и 120
0,025
0,686
VTNC_VDTVDPPYPR_vs_CRIS3_YEDLYSNCK
150 и 79
0,034
0,676
F13B_GDTYPAELYITGSILR_vs_CRIS3_AVSPPAR
84 и 78
0,014
0,705
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,006
0,728
F13B_GDTYPAELYITGSILR_vs_CRIS3_YEDLYSNCK
84 и 79
0,010
0,713
Таблица 55: эффективность классификации по обращениям, недели 19, 20 и 21
AUROC обращения для недель беременности от 19 0/7 до 21 6/7 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, без BMI-стратификации.
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
A2GL_DLLLPQPDLR_vs_IBP3_FLNVLSPR
34 и 99
0,021
0,602
ENPP2_TYLHTYESEI_vs_LYAM1_SYYWIGIR
83 и 120
0,008
0,618
A2GL_DLLLPQPDLR_vs_IBP3_YGQPLPGYTTK
34 и 100
0,013
0,610
ENPP2_TYLHTYESEI_vs_PGRP2_AGLLRPDYALLGHR
83 и 126
0,013
0,610
A2GL_DLLLPQPDLR_vs_IGF2_GIVEECCFR
34 и 103
0,002
0,639
ENPP2_TYLHTYESEI_vs_SHBG_IALGGLLFPASNLR
83 и 18
0,004
0,627
A2GL_DLLLPQPDLR_vs_PRG2_WNFAYWAAHQPWSR
34 и 129
0,019
0,604
ENPP2_TYLHTYESEI_vs_TENX_LNWEAPPGAFDSFLLR
83 и 141
0,008
0,617
A2GL_DLLLPQPDLR_vs_PSG3_VSAPSGTGHLPGLNPL
34 и 134
0,022
0,602
ENPP2_TYLHTYESEI_vs_TENX_LSQLSVTDVTTSSLR
83 и 142
0,014
0,610
A2GL_DLLLPQPDLR_vs_SHBG_IALGGLLFPASNLR
34 и 18
0,003
0,630
F13B_GDTYPAELYITGSILR_vs_IGF2_GIVEECCFR
84 и 103
0,022
0,602
A2GL_DLLLPQPDLR_vs_TENX_LNWEAPPGAFDSFLLR
34 и 141
0,011
0,612
FBLN3_IPSNPSHR_vs_SHBG_IALGGLLFPASNLR
87 и 18
0,017
0,606
AFAM_DADPDTFFAK_vs_IBP3_YGQPLPGYTTK
37 и 100
0,022
0,602
FETUA_FSVVYAK_vs_IBP3_FLNVLSPR
88 и 99
0,012
0,611
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,009
0,616
FETUA_FSVVYAK_vs_IBP3_YGQPLPGYTTK
88 и 100
0,006
0,622
AFAM_HFQNLGK_vs_IBP3_FLNVLSPR
38 и 99
0,018
0,605
FETUA_FSVVYAK_vs_IGF2_GIVEECCFR
88 и 103
0,001
0,654
AFAM_HFQNLGK_vs_IBP3_YGQPLPGYTTK
38 и 100
0,003
0,631
FETUA_FSVVYAK_vs_SHBG_IALGGLLFPASNLR
88 и 18
0,011
0,613
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,001
0,642
FETUA_FSVVYAK_vs_TENX_LNWEAPPGAFDSFLLR
88 и 141
0,007
0,620
APOC3_GWVTDGFSSLK_vs_ALS_IRPHTFTGLSGLR
47 и 40
0,001
0,648
FETUA_FSVVYAK_vs_TENX_LSQLSVTDVTTSSLR
88 и 142
0,013
0,610
APOC3_GWVTDGFSSLK_vs_C163A_INPASLDK
47 и 54
0,002
0,634
FETUA_HTLNQIDEVK_vs_IBP3_YGQPLPGYTTK
89 и 100
0,017
0,606
APOC3_GWVTDGFSSLK_vs_CHL1_VIAVNEVGR
47 и 66
0,009
0,617
FETUA_HTLNQIDEVK_vs_IGF2_GIVEECCFR
89 и 103
0,002
0,636
APOC3_GWVTDGFSSLK_vs_CRIS3_AVSPPAR
47 и 78
0,003
0,631
FETUA_HTLNQIDEVK_vs_SHBG_IALGGLLFPASNLR
89 и 18
0,007
0,619
APOC3_GWVTDGFSSLK_vs_CRIS3_YEDLYSNCK
47 и 79
0,002
0,635
FETUA_HTLNQIDEVK_vs_TENX_LNWEAPPGAFDSFLLR
89 и 141
0,005
0,625
APOC3_GWVTDGFSSLK_vs_CSH_AHQLAIDTYQEFEETYIPK
47 и 80
0,003
0,634
FETUA_HTLNQIDEVK_vs_TENX_LSQLSVTDVTTSSLR
89 и 142
0,010
0,615
APOC3_GWVTDGFSSLK_vs_CSH_ISLLLIESWLEPVR
47 и 81
0,005
0,624
HABP2_FLNWIK_vs_IGF2_GIVEECCFR
92 и 103
0,010
0,614
APOC3_GWVTDGFSSLK_vs_FBLN1_TGYYFDGISR
47 и 86
0,003
0,631
HABP2_FLNWIK_vs_TENX_LNWEAPPGAFDSFLLR
92 и 141
0,022
0,602
APOC3_GWVTDGFSSLK_vs_IBP1_VVESLAK
47 и 97
0,023
0,601
HEMO_NFPSPVDAAFR_vs_IBP3_YGQPLPGYTTK
93 и 100
0,018
0,605
APOC3_GWVTDGFSSLK_vs_IBP2_LIQGAPTIR
47 и 98
0,012
0,611
HEMO_NFPSPVDAAFR_vs_IGF2_GIVEECCFR
93 и 103
0,003
0,632
APOC3_GWVTDGFSSLK_vs_IBP3_FLNVLSPR
47 и 99
0,000
0,666
HEMO_NFPSPVDAAFR_vs_SHBG_IALGGLLFPASNLR
93 и 18
0,005
0,625
APOC3_GWVTDGFSSLK_vs_IBP3_YGQPLPGYTTK
47 и 100
0,000
0,662
HEMO_NFPSPVDAAFR_vs_TENX_LNWEAPPGAFDSFLLR
93 и 141
0,012
0,611
APOC3_GWVTDGFSSLK_vs_IGF2_GIVEECCFR
47 и 103
0,000
0,675
IBP4_QCHPALDGQR_vs_ALS_IRPHTFTGLSGLR
2 и 40
0,007
0,619
APOC3_GWVTDGFSSLK_vs_ITIH4_ILDDLSPR
47 и 112
0,001
0,646
IBP4_QCHPALDGQR_vs_CHL1_VIAVNEVGR
2 и 66
0,023
0,601
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,000
0,658
IBP4_QCHPALDGQR_vs_CRIS3_AVSPPAR
2 и 78
0,004
0,628
APOC3_GWVTDGFSSLK_vs_NCAM1_GLGEISAASEFK
47 и 121
0,003
0,630
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,002
0,637
APOC3_GWVTDGFSSLK_vs_PGRP2_AGLLRPDYALLGHR
47 и 126
0,001
0,652
IBP4_QCHPALDGQR_vs_IBP3_FLNVLSPR
2 и 99
0,002
0,635
APOC3_GWVTDGFSSLK_vs_PRG2_WNFAYWAAHQPWSR
47 и 129
0,003
0,632
IBP4_QCHPALDGQR_vs_IBP3_YGQPLPGYTTK
2 и 100
0,001
0,648
APOC3_GWVTDGFSSLK_vs_PSG1_FQLPGQK
47 и 131
0,016
0,607
IBP4_QCHPALDGQR_vs_IGF2_GIVEECCFR
2 и 103
0,000
0,684
APOC3_GWVTDGFSSLK_vs_PSG3_VSAPSGTGHLPGLNPL
47 и 134
0,001
0,646
IBP4_QCHPALDGQR_vs_ITIH4_ILDDLSPR
2 и 112
0,001
0,641
APOC3_GWVTDGFSSLK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
47 и 135
0,016
0,607
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,001
0,650
APOC3_GWVTDGFSSLK_vs_PSG9_LFIPQITR
47 и 136
0,020
0,603
IBP4_QCHPALDGQR_vs_PGRP2_AGLLRPDYALLGHR
2 и 126
0,000
0,655
APOC3_GWVTDGFSSLK_vs_SHBG_IALGGLLFPASNLR
47 и 18
0,001
0,652
IBP4_QCHPALDGQR_vs_PRG2_WNFAYWAAHQPWSR
2 и 129
0,006
0,622
APOC3_GWVTDGFSSLK_vs_SOM2.CSH_NYGLLYCFR
47 и 138
0,009
0,617
IBP4_QCHPALDGQR_vs_PSG3_VSAPSGTGHLPGLNPL
2 и 134
0,001
0,651
APOC3_GWVTDGFSSLK_vs_SOM2.CSH_SVEGSCGF
47 и 139
0,003
0,631
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,000
0,670
APOC3_GWVTDGFSSLK_vs_SPRL1_VLTHSELAPLR
47 и 140
0,002
0,637
IBP4_QCHPALDGQR_vs_SPRL1_VLTHSELAPLR
2 и 140
0,006
0,622
APOC3_GWVTDGFSSLK_vs_TENX_LNWEAPPGAFDSFLLR
47 и 141
0,001
0,653
IBP4_QCHPALDGQR_vs_TENX_LNWEAPPGAFDSFLLR
2 и 141
0,000
0,661
APOC3_GWVTDGFSSLK_vs_TENX_LSQLSVTDVTTSSLR
47 и 142
0,001
0,651
IBP4_QCHPALDGQR_vs_TENX_LSQLSVTDVTTSSLR
2 и 142
0,001
0,642
APOC3_GWVTDGFSSLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
47 и 144
0,001
0,642
IBP4_QCHPALDGQR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
2 и 144
0,003
0,634
APOC3_GWVTDGFSSLK_vs_VTDB_ELPEHTVK
47 и 147
0,001
0,646
IBP4_QCHPALDGQR_vs_VTDB_ELPEHTVK
2 и 147
0,003
0,632
APOH_ATVVYQGER_vs_IBP3_FLNVLSPR
48 и 99
0,010
0,614
IBP6_GAQTLYVPNCDHR_vs_IGF2_GIVEECCFR
101 и 103
0,008
0,617
APOH_ATVVYQGER_vs_IBP3_YGQPLPGYTTK
48 и 100
0,003
0,631
IBP6_GAQTLYVPNCDHR_vs_SHBG_IALGGLLFPASNLR
101 и 18
0,020
0,604
APOH_ATVVYQGER_vs_IGF2_GIVEECCFR
48 и 103
0,000
0,657
IBP6_HLDSVLQQLQTEVYR_vs_IGF2_GIVEECCFR
102 и 103
0,010
0,615
APOH_ATVVYQGER_vs_LYAM1_SYYWIGIR
48 и 120
0,019
0,604
IBP6_HLDSVLQQLQTEVYR_vs_SHBG_IALGGLLFPASNLR
102 и 18
0,013
0,610
APOH_ATVVYQGER_vs_PGRP2_AGLLRPDYALLGHR
48 и 126
0,018
0,605
INHBC_LDFHFSSDR_vs_ALS_IRPHTFTGLSGLR
107 и 40
0,015
0,608
APOH_ATVVYQGER_vs_PSG3_VSAPSGTGHLPGLNPL
48 и 134
0,024
0,600
INHBC_LDFHFSSDR_vs_CRIS3_YEDLYSNCK
107 и 79
0,023
0,601
APOH_ATVVYQGER_vs_SHBG_IALGGLLFPASNLR
48 и 18
0,007
0,620
INHBC_LDFHFSSDR_vs_IBP3_FLNVLSPR
107 и 99
0,003
0,631
APOH_ATVVYQGER_vs_TENX_LNWEAPPGAFDSFLLR
48 и 141
0,004
0,627
INHBC_LDFHFSSDR_vs_IBP3_YGQPLPGYTTK
107 и 100
0,002
0,638
APOH_ATVVYQGER_vs_TENX_LSQLSVTDVTTSSLR
48 и 142
0,006
0,623
INHBC_LDFHFSSDR_vs_IGF2_GIVEECCFR
107 и 103
0,000
0,664
B2MG_VEHSDLSFSK_vs_IGF2_GIVEECCFR
50 и 103
0,003
0,630
INHBC_LDFHFSSDR_vs_ITIH4_ILDDLSPR
107 и 112
0,013
0,610
B2MG_VEHSDLSFSK_vs_LYAM1_SYYWIGIR
50 и 120
0,012
0,611
INHBC_LDFHFSSDR_vs_LYAM1_SYYWIGIR
107 и 120
0,007
0,620
B2MG_VEHSDLSFSK_vs_SHBG_IALGGLLFPASNLR
50 и 18
0,008
0,617
INHBC_LDFHFSSDR_vs_PGRP2_AGLLRPDYALLGHR
107 и 126
0,004
0,628
B2MG_VEHSDLSFSK_vs_TENX_LNWEAPPGAFDSFLLR
50 и 141
0,013
0,610
INHBC_LDFHFSSDR_vs_PSG3_VSAPSGTGHLPGLNPL
107 и 134
0,007
0,619
B2MG_VEHSDLSFSK_vs_TENX_LSQLSVTDVTTSSLR
50 и 142
0,014
0,609
INHBC_LDFHFSSDR_vs_SHBG_IALGGLLFPASNLR
107 и 18
0,002
0,635
B2MG_VNHVTLSQPK_vs_IBP3_FLNVLSPR
51 и 99
0,013
0,610
INHBC_LDFHFSSDR_vs_SPRL1_VLTHSELAPLR
107 и 140
0,019
0,604
B2MG_VNHVTLSQPK_vs_IBP3_YGQPLPGYTTK
51 и 100
0,010
0,614
INHBC_LDFHFSSDR_vs_TENX_LNWEAPPGAFDSFLLR
107 и 141
0,001
0,643
B2MG_VNHVTLSQPK_vs_IGF2_GIVEECCFR
51 и 103
0,000
0,658
INHBC_LDFHFSSDR_vs_TENX_LSQLSVTDVTTSSLR
107 и 142
0,002
0,636
B2MG_VNHVTLSQPK_vs_LYAM1_SYYWIGIR
51 и 120
0,006
0,622
INHBC_LDFHFSSDR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
107 и 144
0,013
0,611
B2MG_VNHVTLSQPK_vs_PGRP2_AGLLRPDYALLGHR
51 и 126
0,009
0,615
INHBC_LDFHFSSDR_vs_VTDB_ELPEHTVK
107 и 147
0,010
0,614
B2MG_VNHVTLSQPK_vs_SHBG_IALGGLLFPASNLR
51 и 18
0,003
0,634
ITIH3_ALDLSLK_vs_IGF2_GIVEECCFR
111 и 103
0,015
0,608
B2MG_VNHVTLSQPK_vs_TENX_LNWEAPPGAFDSFLLR
51 и 141
0,006
0,621
ITIH3_ALDLSLK_vs_LYAM1_SYYWIGIR
111 и 120
0,022
0,602
B2MG_VNHVTLSQPK_vs_TENX_LSQLSVTDVTTSSLR
51 и 142
0,008
0,618
ITIH3_ALDLSLK_vs_PGRP2_AGLLRPDYALLGHR
111 и 126
0,012
0,611
C1QB_VPGLYYFTYHASSR_vs_IGF2_GIVEECCFR
55 и 103
0,004
0,627
ITIH3_ALDLSLK_vs_SHBG_IALGGLLFPASNLR
111 и 18
0,002
0,636
C1QB_VPGLYYFTYHASSR_vs_SHBG_IALGGLLFPASNLR
55 и 18
0,011
0,613
ITIH3_ALDLSLK_vs_TENX_LNWEAPPGAFDSFLLR
111 и 141
0,014
0,609
C1QB_VPGLYYFTYHASSR_vs_TENX_LNWEAPPGAFDSFLLR
55 и 141
0,012
0,612
KNG1_DIPTNSPELEETLTHTITK_vs_IGF2_GIVEECCFR
116 и 103
0,002
0,637
C1QB_VPGLYYFTYHASSR_vs_TENX_LSQLSVTDVTTSSLR
55 и 142
0,020
0,603
KNG1_DIPTNSPELEETLTHTITK_vs_LYAM1_SYYWIGIR
116 и 120
0,010
0,615
CATD_VGFAEAAR_vs_CRIS3_YEDLYSNCK
57 и 79
0,018
0,605
KNG1_DIPTNSPELEETLTHTITK_vs_SHBG_IALGGLLFPASNLR
116 и 18
0,015
0,608
CATD_VGFAEAAR_vs_IBP3_FLNVLSPR
57 и 99
0,011
0,613
KNG1_DIPTNSPELEETLTHTITK_vs_TENX_LNWEAPPGAFDSFLLR
116 и 141
0,007
0,619
CATD_VGFAEAAR_vs_IBP3_YGQPLPGYTTK
57 и 100
0,012
0,611
KNG1_DIPTNSPELEETLTHTITK_vs_TENX_LSQLSVTDVTTSSLR
116 и 142
0,013
0,611
CATD_VGFAEAAR_vs_IGF2_GIVEECCFR
57 и 103
0,001
0,645
KNG1_QVVAGLNFR_vs_IBP3_YGQPLPGYTTK
117 и 100
0,020
0,603
CATD_VGFAEAAR_vs_LYAM1_SYYWIGIR
57 и 120
0,011
0,613
KNG1_QVVAGLNFR_vs_IGF2_GIVEECCFR
117 и 103
0,003
0,630
CATD_VGFAEAAR_vs_PGRP2_AGLLRPDYALLGHR
57 и 126
0,011
0,613
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,010
0,615
CATD_VGFAEAAR_vs_PRG2_WNFAYWAAHQPWSR
57 и 129
0,007
0,619
KNG1_QVVAGLNFR_vs_PGRP2_AGLLRPDYALLGHR
117 и 126
0,020
0,603
CATD_VGFAEAAR_vs_PSG3_VSAPSGTGHLPGLNPL
57 и 134
0,010
0,614
KNG1_QVVAGLNFR_vs_SHBG_IALGGLLFPASNLR
117 и 18
0,007
0,619
CATD_VGFAEAAR_vs_SHBG_IALGGLLFPASNLR
57 и 18
0,006
0,621
KNG1_QVVAGLNFR_vs_TENX_LNWEAPPGAFDSFLLR
117 и 141
0,012
0,612
CATD_VGFAEAAR_vs_TENX_LNWEAPPGAFDSFLLR
57 и 141
0,002
0,636
KNG1_QVVAGLNFR_vs_TENX_LSQLSVTDVTTSSLR
117 и 142
0,023
0,601
CATD_VGFAEAAR_vs_TENX_LSQLSVTDVTTSSLR
57 и 142
0,003
0,631
LBP_ITGFLKPGK_vs_CRIS3_AVSPPAR
118 и 78
0,016
0,606
CATD_VSTLPAITLK_vs_IBP3_FLNVLSPR
58 и 99
0,014
0,609
LBP_ITGFLKPGK_vs_CRIS3_YEDLYSNCK
118 и 79
0,010
0,615
CATD_VSTLPAITLK_vs_IBP3_YGQPLPGYTTK
58 и 100
0,015
0,608
LBP_ITGFLKPGK_vs_IBP3_FLNVLSPR
118 и 99
0,015
0,608
CATD_VSTLPAITLK_vs_IGF2_GIVEECCFR
58 и 103
0,002
0,640
LBP_ITGFLKPGK_vs_IBP3_YGQPLPGYTTK
118 и 100
0,010
0,614
CATD_VSTLPAITLK_vs_PGRP2_AGLLRPDYALLGHR
58 и 126
0,016
0,607
LBP_ITGFLKPGK_vs_IGF2_GIVEECCFR
118 и 103
0,002
0,638
CATD_VSTLPAITLK_vs_PRG2_WNFAYWAAHQPWSR
58 и 129
0,023
0,601
LBP_ITGFLKPGK_vs_LYAM1_SYYWIGIR
118 и 120
0,012
0,612
CATD_VSTLPAITLK_vs_PSG3_VSAPSGTGHLPGLNPL
58 и 134
0,022
0,602
LBP_ITGFLKPGK_vs_PGRP2_AGLLRPDYALLGHR
118 и 126
0,012
0,611
CATD_VSTLPAITLK_vs_SHBG_IALGGLLFPASNLR
58 и 18
0,008
0,618
LBP_ITGFLKPGK_vs_PRG2_WNFAYWAAHQPWSR
118 и 129
0,022
0,601
CATD_VSTLPAITLK_vs_TENX_LNWEAPPGAFDSFLLR
58 и 141
0,004
0,628
LBP_ITGFLKPGK_vs_PSG3_VSAPSGTGHLPGLNPL
118 и 134
0,004
0,627
CATD_VSTLPAITLK_vs_TENX_LSQLSVTDVTTSSLR
58 и 142
0,007
0,620
LBP_ITGFLKPGK_vs_SHBG_IALGGLLFPASNLR
118 и 18
0,004
0,628
CD14_LTVGAAQVPAQLLVGALR_vs_IBP3_FLNVLSPR
61 и 99
0,009
0,615
LBP_ITGFLKPGK_vs_SPRL1_VLTHSELAPLR
118 и 140
0,024
0,600
CD14_LTVGAAQVPAQLLVGALR_vs_IBP3_YGQPLPGYTTK
61 и 100
0,011
0,612
LBP_ITGFLKPGK_vs_TENX_LNWEAPPGAFDSFLLR
118 и 141
0,010
0,614
CD14_LTVGAAQVPAQLLVGALR_vs_IGF2_GIVEECCFR
61 и 103
0,001
0,649
LBP_ITGFLKPGK_vs_TENX_LSQLSVTDVTTSSLR
118 и 142
0,018
0,605
CD14_LTVGAAQVPAQLLVGALR_vs_LYAM1_SYYWIGIR
61 и 120
0,015
0,608
LBP_ITLPDFTGDLR_vs_ALS_IRPHTFTGLSGLR
119 и 40
0,013
0,611
CD14_LTVGAAQVPAQLLVGALR_vs_PGRP2_AGLLRPDYALLGHR
61 и 126
0,024
0,600
LBP_ITLPDFTGDLR_vs_C163A_INPASLDK
119 и 54
0,017
0,606
CD14_LTVGAAQVPAQLLVGALR_vs_SHBG_IALGGLLFPASNLR
61 и 18
0,007
0,619
LBP_ITLPDFTGDLR_vs_CHL1_VIAVNEVGR
119 и 66
0,024
0,600
CD14_LTVGAAQVPAQLLVGALR_vs_TENX_LNWEAPPGAFDSFLLR
61 и 141
0,005
0,623
LBP_ITLPDFTGDLR_vs_CRIS3_AVSPPAR
119 и 78
0,005
0,624
CD14_LTVGAAQVPAQLLVGALR_vs_TENX_LSQLSVTDVTTSSLR
61 и 142
0,012
0,611
LBP_ITLPDFTGDLR_vs_CRIS3_YEDLYSNCK
119 и 79
0,003
0,633
CD14_SWLAELQQWLKPGLK_vs_IGF2_GIVEECCFR
62 и 103
0,003
0,630
LBP_ITLPDFTGDLR_vs_IBP2_LIQGAPTIR
119 и 98
0,024
0,600
CD14_SWLAELQQWLKPGLK_vs_LYAM1_SYYWIGIR
62 и 120
0,022
0,602
LBP_ITLPDFTGDLR_vs_IBP3_FLNVLSPR
119 и 99
0,003
0,630
CD14_SWLAELQQWLKPGLK_vs_SHBG_IALGGLLFPASNLR
62 и 18
0,011
0,613
LBP_ITLPDFTGDLR_vs_IBP3_YGQPLPGYTTK
119 и 100
0,002
0,638
CD14_SWLAELQQWLKPGLK_vs_TENX_LNWEAPPGAFDSFLLR
62 и 141
0,009
0,615
LBP_ITLPDFTGDLR_vs_IGF2_GIVEECCFR
119 и 103
0,000
0,655
CD14_SWLAELQQWLKPGLK_vs_TENX_LSQLSVTDVTTSSLR
62 и 142
0,024
0,600
LBP_ITLPDFTGDLR_vs_ITIH4_ILDDLSPR
119 и 112
0,006
0,621
CFAB_YGLVTYATYPK_vs_IBP3_YGQPLPGYTTK
64 и 100
0,017
0,606
LBP_ITLPDFTGDLR_vs_LYAM1_SYYWIGIR
119 и 120
0,003
0,632
CFAB_YGLVTYATYPK_vs_IGF2_GIVEECCFR
64 и 103
0,003
0,634
LBP_ITLPDFTGDLR_vs_PGRP2_AGLLRPDYALLGHR
119 и 126
0,003
0,630
CFAB_YGLVTYATYPK_vs_SHBG_IALGGLLFPASNLR
64 и 18
0,015
0,608
LBP_ITLPDFTGDLR_vs_PRG2_WNFAYWAAHQPWSR
119 и 129
0,009
0,615
CO5_TLLPVSKPEIR_vs_IGF2_GIVEECCFR
70 и 103
0,006
0,622
LBP_ITLPDFTGDLR_vs_PSG3_VSAPSGTGHLPGLNPL
119 и 134
0,002
0,640
CO5_TLLPVSKPEIR_vs_SHBG_IALGGLLFPASNLR
70 и 18
0,016
0,607
LBP_ITLPDFTGDLR_vs_SHBG_IALGGLLFPASNLR
119 и 18
0,001
0,645
CO5_TLLPVSKPEIR_vs_TENX_LNWEAPPGAFDSFLLR
70 и 141
0,016
0,607
LBP_ITLPDFTGDLR_vs_SOM2.CSH_SVEGSCGF
119 и 139
0,023
0,601
CO5_VFQFLEK_vs_IGF2_GIVEECCFR
71 и 103
0,010
0,614
LBP_ITLPDFTGDLR_vs_SPRL1_VLTHSELAPLR
119 и 140
0,009
0,616
CO5_VFQFLEK_vs_SHBG_IALGGLLFPASNLR
71 и 18
0,017
0,606
LBP_ITLPDFTGDLR_vs_TENX_LNWEAPPGAFDSFLLR
119 и 141
0,002
0,634
CO5_VFQFLEK_vs_TENX_LNWEAPPGAFDSFLLR
71 и 141
0,015
0,608
LBP_ITLPDFTGDLR_vs_TENX_LSQLSVTDVTTSSLR
119 и 142
0,005
0,625
CO6_ALNHLPLEYNSALYSR_vs_IGF2_GIVEECCFR
72 и 103
0,020
0,603
LBP_ITLPDFTGDLR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
119 и 144
0,011
0,613
CO8A_SLLQPNK_vs_ALS_IRPHTFTGLSGLR
74 и 40
0,023
0,601
LBP_ITLPDFTGDLR_vs_VTDB_ELPEHTVK
119 и 147
0,007
0,621
CO8A_SLLQPNK_vs_IBP3_FLNVLSPR
74 и 99
0,012
0,611
PEDF_LQSLFDSPDFSK_vs_IGF2_GIVEECCFR
124 и 103
0,008
0,617
CO8A_SLLQPNK_vs_IBP3_YGQPLPGYTTK
74 и 100
0,006
0,623
PEDF_LQSLFDSPDFSK_vs_TENX_LNWEAPPGAFDSFLLR
124 и 141
0,012
0,612
CO8A_SLLQPNK_vs_IGF2_GIVEECCFR
74 и 103
0,000
0,656
PEDF_LQSLFDSPDFSK_vs_TENX_LSQLSVTDVTTSSLR
124 и 142
0,013
0,610
CO8A_SLLQPNK_vs_LYAM1_SYYWIGIR
74 и 120
0,021
0,602
PEDF_TVQAVLTVPK_vs_IGF2_GIVEECCFR
125 и 103
0,003
0,630
CO8A_SLLQPNK_vs_PGRP2_AGLLRPDYALLGHR
74 и 126
0,018
0,605
PEDF_TVQAVLTVPK_vs_TENX_LNWEAPPGAFDSFLLR
125 и 141
0,014
0,609
CO8A_SLLQPNK_vs_SHBG_IALGGLLFPASNLR
74 и 18
0,010
0,615
PEDF_TVQAVLTVPK_vs_TENX_LSQLSVTDVTTSSLR
125 и 142
0,018
0,604
CO8A_SLLQPNK_vs_TENX_LNWEAPPGAFDSFLLR
74 и 141
0,005
0,624
PTGDS_GPGEDFR_vs_IGF2_GIVEECCFR
137 и 103
0,008
0,618
CO8A_SLLQPNK_vs_TENX_LSQLSVTDVTTSSLR
74 и 142
0,005
0,624
PTGDS_GPGEDFR_vs_SHBG_IALGGLLFPASNLR
137 и 18
0,016
0,607
CO8B_QALEEFQK_vs_ALS_IRPHTFTGLSGLR
76 и 40
0,012
0,612
PTGDS_GPGEDFR_vs_TENX_LNWEAPPGAFDSFLLR
137 и 141
0,013
0,610
CO8B_QALEEFQK_vs_IBP3_FLNVLSPR
76 и 99
0,009
0,617
PTGDS_GPGEDFR_vs_TENX_LSQLSVTDVTTSSLR
137 и 142
0,016
0,607
CO8B_QALEEFQK_vs_IBP3_YGQPLPGYTTK
76 и 100
0,005
0,624
THBG_AVLHIGEK_vs_IGF2_GIVEECCFR
143 и 103
0,011
0,612
CO8B_QALEEFQK_vs_IGF2_GIVEECCFR
76 и 103
0,000
0,663
THBG_AVLHIGEK_vs_SHBG_IALGGLLFPASNLR
143 и 18
0,013
0,610
CO8B_QALEEFQK_vs_LYAM1_SYYWIGIR
76 и 120
0,015
0,608
VTNC_GQYCYELDEK_vs_IBP3_FLNVLSPR
149 и 99
0,016
0,607
CO8B_QALEEFQK_vs_PGRP2_AGLLRPDYALLGHR
76 и 126
0,019
0,604
VTNC_GQYCYELDEK_vs_IBP3_YGQPLPGYTTK
149 и 100
0,006
0,621
CO8B_QALEEFQK_vs_PSG3_VSAPSGTGHLPGLNPL
76 и 134
0,020
0,603
VTNC_GQYCYELDEK_vs_IGF2_GIVEECCFR
149 и 103
0,001
0,641
CO8B_QALEEFQK_vs_SHBG_IALGGLLFPASNLR
76 и 18
0,011
0,613
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,015
0,608
CO8B_QALEEFQK_vs_TENX_LNWEAPPGAFDSFLLR
76 и 141
0,003
0,632
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,002
0,639
CO8B_QALEEFQK_vs_TENX_LSQLSVTDVTTSSLR
76 и 142
0,002
0,637
VTNC_GQYCYELDEK_vs_TENX_LNWEAPPGAFDSFLLR
149 и 141
0,012
0,612
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_IBP3_FLNVLSPR
82 и 99
0,016
0,607
VTNC_GQYCYELDEK_vs_TENX_LSQLSVTDVTTSSLR
149 и 142
0,018
0,605
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_IBP3_YGQPLPGYTTK
82 и 100
0,013
0,610
VTNC_VDTVDPPYPR_vs_IBP3_FLNVLSPR
150 и 99
0,007
0,619
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_IGF2_GIVEECCFR
82 и 103
0,004
0,629
VTNC_VDTVDPPYPR_vs_IBP3_YGQPLPGYTTK
150 и 100
0,004
0,628
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_LYAM1_SYYWIGIR
82 и 120
0,011
0,613
VTNC_VDTVDPPYPR_vs_IGF2_GIVEECCFR
150 и 103
0,001
0,649
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PGRP2_AGLLRPDYALLGHR
82 и 126
0,017
0,606
VTNC_VDTVDPPYPR_vs_ITIH4_ILDDLSPR
150 и 112
0,016
0,606
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PRG2_WNFAYWAAHQPWSR
82 и 129
0,024
0,600
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,005
0,625
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_SHBG_IALGGLLFPASNLR
82 и 18
0,003
0,633
VTNC_VDTVDPPYPR_vs_PGRP2_AGLLRPDYALLGHR
150 и 126
0,009
0,616
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_TENX_LNWEAPPGAFDSFLLR
82 и 141
0,014
0,609
VTNC_VDTVDPPYPR_vs_PSG3_VSAPSGTGHLPGLNPL
150 и 134
0,021
0,602
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_TENX_LSQLSVTDVTTSSLR
82 и 142
0,019
0,604
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,001
0,644
ENPP2_TYLHTYESEI_vs_IBP3_FLNVLSPR
83 и 99
0,012
0,611
VTNC_VDTVDPPYPR_vs_TENX_LNWEAPPGAFDSFLLR
150 и 141
0,005
0,624
ENPP2_TYLHTYESEI_vs_IBP3_YGQPLPGYTTK
83 и 100
0,012
0,611
VTNC_VDTVDPPYPR_vs_TENX_LSQLSVTDVTTSSLR
150 и 142
0,009
0,615
ENPP2_TYLHTYESEI_vs_IGF2_GIVEECCFR
83 и 103
0,003
0,631
VTNC_VDTVDPPYPR_vs_VTDB_ELPEHTVK
150 и 147
0,008
0,618
Таблица 56: эффективность классификации по обращениям, недели 19, 20 и 21
AUROC обращения для недель беременности от 19 0/7 до 21 6/7 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, с BMI-стратификацией (>22≤37).
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
A2GL_DLLLPQPDLR_vs_CRIS3_AVSPPAR
34 и 78
0,023
0,622
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PGRP2_AGLLRPDYALLGHR
82 и 126
0,017
0,628
A2GL_DLLLPQPDLR_vs_CRIS3_YEDLYSNCK
34 и 79
0,011
0,636
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
82 и 135
0,030
0,617
A2GL_DLLLPQPDLR_vs_IBP3_FLNVLSPR
34 и 99
0,023
0,622
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_SHBG_IALGGLLFPASNLR
82 и 18
0,005
0,652
A2GL_DLLLPQPDLR_vs_IBP3_YGQPLPGYTTK
34 и 100
0,015
0,631
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_SOM2.CSH_SVEGSCGF
82 и 139
0,029
0,617
A2GL_DLLLPQPDLR_vs_IGF2_GIVEECCFR
34 и 103
0,003
0,660
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_TENX_LNWEAPPGAFDSFLLR
82 и 141
0,030
0,617
A2GL_DLLLPQPDLR_vs_LYAM1_SYYWIGIR
34 и 120
0,003
0,658
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_TENX_LSQLSVTDVTTSSLR
82 и 142
0,048
0,606
A2GL_DLLLPQPDLR_vs_PGRP2_AGLLRPDYALLGHR
34 и 126
0,021
0,624
ENPP2_TYLHTYESEI_vs_C163A_INPASLDK
83 и 54
0,047
0,607
A2GL_DLLLPQPDLR_vs_PSG3_VSAPSGTGHLPGLNPL
34 и 134
0,044
0,608
ENPP2_TYLHTYESEI_vs_CRIS3_AVSPPAR
83 и 78
0,050
0,606
A2GL_DLLLPQPDLR_vs_SHBG_IALGGLLFPASNLR
34 и 18
0,012
0,636
ENPP2_TYLHTYESEI_vs_CRIS3_YEDLYSNCK
83 и 79
0,029
0,618
A2GL_DLLLPQPDLR_vs_TENX_LNWEAPPGAFDSFLLR
34 и 141
0,024
0,622
ENPP2_TYLHTYESEI_vs_IBP3_FLNVLSPR
83 и 99
0,032
0,615
AFAM_DADPDTFFAK_vs_IBP3_FLNVLSPR
37 и 99
0,033
0,614
ENPP2_TYLHTYESEI_vs_IBP3_YGQPLPGYTTK
83 и 100
0,022
0,623
AFAM_DADPDTFFAK_vs_IBP3_YGQPLPGYTTK
37 и 100
0,004
0,653
ENPP2_TYLHTYESEI_vs_IGF2_GIVEECCFR
83 и 103
0,014
0,632
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,004
0,656
ENPP2_TYLHTYESEI_vs_LYAM1_SYYWIGIR
83 и 120
0,011
0,637
AFAM_DADPDTFFAK_vs_TENX_LNWEAPPGAFDSFLLR
37 и 141
0,046
0,607
ENPP2_TYLHTYESEI_vs_PGRP2_AGLLRPDYALLGHR
83 и 126
0,036
0,613
AFAM_HFQNLGK_vs_ALS_IRPHTFTGLSGLR
38 и 40
0,035
0,614
ENPP2_TYLHTYESEI_vs_SHBG_IALGGLLFPASNLR
83 и 18
0,010
0,638
AFAM_HFQNLGK_vs_CRIS3_YEDLYSNCK
38 и 79
0,035
0,613
ENPP2_TYLHTYESEI_vs_SOM2.CSH_SVEGSCGF
83 и 139
0,042
0,610
AFAM_HFQNLGK_vs_IBP3_FLNVLSPR
38 и 99
0,004
0,653
ENPP2_TYLHTYESEI_vs_TENX_LNWEAPPGAFDSFLLR
83 и 141
0,038
0,612
AFAM_HFQNLGK_vs_IBP3_YGQPLPGYTTK
38 и 100
0,001
0,683
F13B_GDTYPAELYITGSILR_vs_CRIS3_YEDLYSNCK
84 и 79
0,029
0,617
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,001
0,685
F13B_GDTYPAELYITGSILR_vs_IBP3_YGQPLPGYTTK
84 и 100
0,040
0,610
AFAM_HFQNLGK_vs_LYAM1_SYYWIGIR
38 и 120
0,034
0,614
F13B_GDTYPAELYITGSILR_vs_IGF2_GIVEECCFR
84 и 103
0,018
0,628
AFAM_HFQNLGK_vs_PGRP2_AGLLRPDYALLGHR
38 и 126
0,036
0,613
FETUA_FSVVYAK_vs_CRIS3_AVSPPAR
88 и 78
0,039
0,611
AFAM_HFQNLGK_vs_PSG3_VSAPSGTGHLPGLNPL
38 и 134
0,045
0,608
FETUA_FSVVYAK_vs_CRIS3_YEDLYSNCK
88 и 79
0,015
0,631
AFAM_HFQNLGK_vs_TENX_LNWEAPPGAFDSFLLR
38 и 141
0,028
0,618
FETUA_FSVVYAK_vs_IBP3_FLNVLSPR
88 и 99
0,033
0,615
AFAM_HFQNLGK_vs_TENX_LSQLSVTDVTTSSLR
38 и 142
0,043
0,609
FETUA_FSVVYAK_vs_IBP3_YGQPLPGYTTK
88 и 100
0,013
0,633
ANGT_DPTFIPAPIQAK_vs_CRIS3_AVSPPAR
42 и 78
0,026
0,620
FETUA_FSVVYAK_vs_IGF2_GIVEECCFR
88 и 103
0,002
0,664
ANGT_DPTFIPAPIQAK_vs_CRIS3_YEDLYSNCK
42 и 79
0,017
0,629
FETUA_FSVVYAK_vs_LYAM1_SYYWIGIR
88 и 120
0,038
0,612
ANGT_DPTFIPAPIQAK_vs_IBP3_YGQPLPGYTTK
42 и 100
0,040
0,611
FETUA_FSVVYAK_vs_TENX_LNWEAPPGAFDSFLLR
88 и 141
0,049
0,606
ANGT_DPTFIPAPIQAK_vs_IGF2_GIVEECCFR
42 и 103
0,017
0,628
FETUA_HTLNQIDEVK_vs_CRIS3_YEDLYSNCK
89 и 79
0,027
0,619
ANGT_DPTFIPAPIQAK_vs_SHBG_IALGGLLFPASNLR
42 и 18
0,022
0,623
FETUA_HTLNQIDEVK_vs_IBP3_YGQPLPGYTTK
89 и 100
0,024
0,621
ANGT_DPTFIPAPIQAK_vs_TENX_LNWEAPPGAFDSFLLR
42 и 141
0,037
0,612
FETUA_HTLNQIDEVK_vs_IGF2_GIVEECCFR
89 и 103
0,005
0,652
APOC3_GWVTDGFSSLK_vs_ALS_IRPHTFTGLSGLR
47 и 40
0,026
0,620
FETUA_HTLNQIDEVK_vs_LYAM1_SYYWIGIR
89 и 120
0,048
0,606
APOC3_GWVTDGFSSLK_vs_C163A_INPASLDK
47 и 54
0,018
0,627
FETUA_HTLNQIDEVK_vs_SHBG_IALGGLLFPASNLR
89 и 18
0,042
0,610
APOC3_GWVTDGFSSLK_vs_CRIS3_AVSPPAR
47 и 78
0,009
0,641
FETUA_HTLNQIDEVK_vs_TENX_LNWEAPPGAFDSFLLR
89 и 141
0,035
0,613
APOC3_GWVTDGFSSLK_vs_CRIS3_YEDLYSNCK
47 и 79
0,006
0,649
HABP2_FLNWIK_vs_IBP3_YGQPLPGYTTK
92 и 100
0,041
0,610
APOC3_GWVTDGFSSLK_vs_IBP3_FLNVLSPR
47 и 99
0,010
0,639
HABP2_FLNWIK_vs_IGF2_GIVEECCFR
92 и 103
0,022
0,623
APOC3_GWVTDGFSSLK_vs_IBP3_YGQPLPGYTTK
47 и 100
0,009
0,641
HABP2_FLNWIK_vs_TENX_LNWEAPPGAFDSFLLR
92 и 141
0,046
0,607
APOC3_GWVTDGFSSLK_vs_IGF2_GIVEECCFR
47 и 103
0,005
0,653
HEMO_NFPSPVDAAFR_vs_CRIS3_AVSPPAR
93 и 78
0,044
0,608
APOC3_GWVTDGFSSLK_vs_ITIH4_ILDDLSPR
47 и 112
0,020
0,625
HEMO_NFPSPVDAAFR_vs_CRIS3_YEDLYSNCK
93 и 79
0,024
0,621
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,006
0,649
HEMO_NFPSPVDAAFR_vs_IBP3_YGQPLPGYTTK
93 и 100
0,041
0,610
APOC3_GWVTDGFSSLK_vs_NCAM1_GLGEISAASEFK
47 и 121
0,045
0,608
HEMO_NFPSPVDAAFR_vs_IGF2_GIVEECCFR
93 и 103
0,013
0,633
APOC3_GWVTDGFSSLK_vs_PGRP2_AGLLRPDYALLGHR
47 и 126
0,011
0,637
HEMO_NFPSPVDAAFR_vs_LYAM1_SYYWIGIR
93 и 120
0,032
0,615
APOC3_GWVTDGFSSLK_vs_PSG3_VSAPSGTGHLPGLNPL
47 и 134
0,020
0,625
HEMO_NFPSPVDAAFR_vs_SHBG_IALGGLLFPASNLR
93 и 18
0,028
0,618
APOC3_GWVTDGFSSLK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
47 и 135
0,027
0,619
HLACI_WAAVVVPSGEEQR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
95 и 135
0,037
0,612
APOC3_GWVTDGFSSLK_vs_SHBG_IALGGLLFPASNLR
47 и 18
0,011
0,636
IBP4_QCHPALDGQR_vs_ALS_IRPHTFTGLSGLR
2 и 40
0,009
0,641
APOC3_GWVTDGFSSLK_vs_SOM2.CSH_SVEGSCGF
47 и 139
0,018
0,627
IBP4_QCHPALDGQR_vs_CRIS3_AVSPPAR
2 и 78
0,001
0,682
APOC3_GWVTDGFSSLK_vs_SPRL1_VLTHSELAPLR
47 и 140
0,035
0,613
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,000
0,695
APOC3_GWVTDGFSSLK_vs_TENX_LNWEAPPGAFDSFLLR
47 и 141
0,016
0,630
IBP4_QCHPALDGQR_vs_CSH_AHQLAIDTYQEFEETYIPK
2 и 80
0,031
0,616
APOC3_GWVTDGFSSLK_vs_TENX_LSQLSVTDVTTSSLR
47 и 142
0,030
0,617
IBP4_QCHPALDGQR_vs_IBP2_LIQGAPTIR
2 и 98
0,046
0,607
APOC3_GWVTDGFSSLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
47 и 144
0,030
0,616
IBP4_QCHPALDGQR_vs_IBP3_FLNVLSPR
2 и 99
0,002
0,663
APOC3_GWVTDGFSSLK_vs_VTDB_ELPEHTVK
47 и 147
0,026
0,619
IBP4_QCHPALDGQR_vs_IBP3_YGQPLPGYTTK
2 и 100
0,001
0,684
APOH_ATVVYQGER_vs_CRIS3_AVSPPAR
48 и 78
0,031
0,616
IBP4_QCHPALDGQR_vs_IGF2_GIVEECCFR
2 и 103
0,000
0,708
APOH_ATVVYQGER_vs_CRIS3_YEDLYSNCK
48 и 79
0,015
0,631
IBP4_QCHPALDGQR_vs_ITIH4_ILDDLSPR
2 и 112
0,004
0,657
APOH_ATVVYQGER_vs_IBP3_FLNVLSPR
48 и 99
0,035
0,613
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,000
0,707
APOH_ATVVYQGER_vs_IBP3_YGQPLPGYTTK
48 и 100
0,007
0,645
IBP4_QCHPALDGQR_vs_NCAM1_GLGEISAASEFK
2 и 121
0,025
0,620
APOH_ATVVYQGER_vs_IGF2_GIVEECCFR
48 и 103
0,005
0,652
IBP4_QCHPALDGQR_vs_PGRP2_AGLLRPDYALLGHR
2 и 126
0,001
0,687
APOH_ATVVYQGER_vs_LYAM1_SYYWIGIR
48 и 120
0,022
0,623
IBP4_QCHPALDGQR_vs_PSG3_VSAPSGTGHLPGLNPL
2 и 134
0,002
0,663
APOH_ATVVYQGER_vs_PGRP2_AGLLRPDYALLGHR
48 и 126
0,030
0,616
IBP4_QCHPALDGQR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
2 и 135
0,040
0,610
APOH_ATVVYQGER_vs_SHBG_IALGGLLFPASNLR
48 и 18
0,033
0,615
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,001
0,680
APOH_ATVVYQGER_vs_TENX_LNWEAPPGAFDSFLLR
48 и 141
0,033
0,615
IBP4_QCHPALDGQR_vs_SOM2.CSH_SVEGSCGF
2 и 139
0,014
0,632
B2MG_VEHSDLSFSK_vs_CRIS3_AVSPPAR
50 и 78
0,036
0,613
IBP4_QCHPALDGQR_vs_SPRL1_VLTHSELAPLR
2 и 140
0,007
0,644
B2MG_VEHSDLSFSK_vs_CRIS3_YEDLYSNCK
50 и 79
0,015
0,631
IBP4_QCHPALDGQR_vs_TENX_LNWEAPPGAFDSFLLR
2 и 141
0,001
0,675
B2MG_VEHSDLSFSK_vs_IBP3_YGQPLPGYTTK
50 и 100
0,022
0,623
IBP4_QCHPALDGQR_vs_TENX_LSQLSVTDVTTSSLR
2 и 142
0,009
0,641
B2MG_VEHSDLSFSK_vs_IGF2_GIVEECCFR
50 и 103
0,007
0,644
IBP4_QCHPALDGQR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
2 и 144
0,002
0,663
B2MG_VEHSDLSFSK_vs_LYAM1_SYYWIGIR
50 и 120
0,007
0,645
IBP4_QCHPALDGQR_vs_VTDB_ELPEHTVK
2 и 147
0,003
0,660
B2MG_VEHSDLSFSK_vs_SHBG_IALGGLLFPASNLR
50 и 18
0,036
0,613
INHBC_LDFHFSSDR_vs_ALS_IRPHTFTGLSGLR
107 и 40
0,004
0,654
B2MG_VEHSDLSFSK_vs_TENX_LNWEAPPGAFDSFLLR
50 и 141
0,035
0,613
INHBC_LDFHFSSDR_vs_CRIS3_AVSPPAR
107 и 78
0,012
0,635
B2MG_VNHVTLSQPK_vs_CRIS3_AVSPPAR
51 и 78
0,019
0,627
INHBC_LDFHFSSDR_vs_CRIS3_YEDLYSNCK
107 и 79
0,006
0,649
B2MG_VNHVTLSQPK_vs_CRIS3_YEDLYSNCK
51 и 79
0,009
0,641
INHBC_LDFHFSSDR_vs_CSH_AHQLAIDTYQEFEETYIPK
107 и 80
0,048
0,606
B2MG_VNHVTLSQPK_vs_IBP3_FLNVLSPR
51 и 99
0,019
0,626
INHBC_LDFHFSSDR_vs_IBP3_FLNVLSPR
107 и 99
0,001
0,673
B2MG_VNHVTLSQPK_vs_IBP3_YGQPLPGYTTK
51 и 100
0,011
0,637
INHBC_LDFHFSSDR_vs_IBP3_YGQPLPGYTTK
107 и 100
0,001
0,685
B2MG_VNHVTLSQPK_vs_IGF2_GIVEECCFR
51 и 103
0,002
0,669
INHBC_LDFHFSSDR_vs_IGF2_GIVEECCFR
107 и 103
0,000
0,704
B2MG_VNHVTLSQPK_vs_LYAM1_SYYWIGIR
51 и 120
0,005
0,651
INHBC_LDFHFSSDR_vs_ITIH4_ILDDLSPR
107 и 112
0,014
0,633
B2MG_VNHVTLSQPK_vs_PGRP2_AGLLRPDYALLGHR
51 и 126
0,015
0,630
INHBC_LDFHFSSDR_vs_LYAM1_SYYWIGIR
107 и 120
0,005
0,649
B2MG_VNHVTLSQPK_vs_SHBG_IALGGLLFPASNLR
51 и 18
0,013
0,634
INHBC_LDFHFSSDR_vs_NCAM1_GLGEISAASEFK
107 и 121
0,033
0,614
B2MG_VNHVTLSQPK_vs_TENX_LNWEAPPGAFDSFLLR
51 и 141
0,031
0,616
INHBC_LDFHFSSDR_vs_PGRP2_AGLLRPDYALLGHR
107 и 126
0,005
0,650
B2MG_VNHVTLSQPK_vs_TENX_LSQLSVTDVTTSSLR
51 и 142
0,042
0,609
INHBC_LDFHFSSDR_vs_PSG3_VSAPSGTGHLPGLNPL
107 и 134
0,005
0,651
BGH3_LTLLAPLNSVFK_vs_CRIS3_AVSPPAR
52 и 78
0,038
0,611
INHBC_LDFHFSSDR_vs_SHBG_IALGGLLFPASNLR
107 и 18
0,003
0,657
BGH3_LTLLAPLNSVFK_vs_CRIS3_YEDLYSNCK
52 и 79
0,012
0,635
INHBC_LDFHFSSDR_vs_SOM2.CSH_SVEGSCGF
107 и 139
0,020
0,625
BGH3_LTLLAPLNSVFK_vs_IBP3_YGQPLPGYTTK
52 и 100
0,042
0,609
INHBC_LDFHFSSDR_vs_SPRL1_VLTHSELAPLR
107 и 140
0,013
0,633
BGH3_LTLLAPLNSVFK_vs_IGF2_GIVEECCFR
52 и 103
0,025
0,620
INHBC_LDFHFSSDR_vs_TENX_LNWEAPPGAFDSFLLR
107 и 141
0,001
0,681
BGH3_LTLLAPLNSVFK_vs_LYAM1_SYYWIGIR
52 и 120
0,023
0,622
INHBC_LDFHFSSDR_vs_TENX_LSQLSVTDVTTSSLR
107 и 142
0,002
0,665
BGH3_LTLLAPLNSVFK_vs_TENX_LNWEAPPGAFDSFLLR
52 и 141
0,030
0,617
INHBC_LDFHFSSDR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
107 и 144
0,018
0,627
C1QB_VPGLYYFTYHASSR_vs_IGF2_GIVEECCFR
55 и 103
0,017
0,628
INHBC_LDFHFSSDR_vs_VTDB_ELPEHTVK
107 и 147
0,004
0,653
C1QB_VPGLYYFTYHASSR_vs_TENX_LNWEAPPGAFDSFLLR
55 и 141
0,049
0,606
ITIH3_ALDLSLK_vs_CRIS3_AVSPPAR
111 и 78
0,019
0,626
CATD_VGFAEAAR_vs_CRIS3_AVSPPAR
57 и 78
0,024
0,622
ITIH3_ALDLSLK_vs_CRIS3_YEDLYSNCK
111 и 79
0,012
0,634
CATD_VGFAEAAR_vs_CRIS3_YEDLYSNCK
57 и 79
0,013
0,634
ITIH3_ALDLSLK_vs_IGF2_GIVEECCFR
111 и 103
0,041
0,610
CATD_VGFAEAAR_vs_IBP3_FLNVLSPR
57 и 99
0,032
0,616
ITIH3_ALDLSLK_vs_LYAM1_SYYWIGIR
111 и 120
0,013
0,633
CATD_VGFAEAAR_vs_IBP3_YGQPLPGYTTK
57 и 100
0,022
0,623
ITIH3_ALDLSLK_vs_PGRP2_AGLLRPDYALLGHR
111 и 126
0,027
0,619
CATD_VGFAEAAR_vs_IGF2_GIVEECCFR
57 и 103
0,010
0,639
ITIH3_ALDLSLK_vs_SHBG_IALGGLLFPASNLR
111 и 18
0,015
0,631
CATD_VGFAEAAR_vs_LYAM1_SYYWIGIR
57 и 120
0,022
0,623
KNG1_DIPTNSPELEETLTHTITK_vs_CRIS3_AVSPPAR
116 и 78
0,043
0,609
CATD_VGFAEAAR_vs_PGRP2_AGLLRPDYALLGHR
57 и 126
0,042
0,609
KNG1_DIPTNSPELEETLTHTITK_vs_CRIS3_YEDLYSNCK
116 и 79
0,017
0,628
CATD_VGFAEAAR_vs_SHBG_IALGGLLFPASNLR
57 и 18
0,041
0,610
KNG1_DIPTNSPELEETLTHTITK_vs_IGF2_GIVEECCFR
116 и 103
0,013
0,634
CATD_VGFAEAAR_vs_TENX_LNWEAPPGAFDSFLLR
57 и 141
0,011
0,637
KNG1_DIPTNSPELEETLTHTITK_vs_LYAM1_SYYWIGIR
116 и 120
0,015
0,631
CATD_VGFAEAAR_vs_TENX_LSQLSVTDVTTSSLR
57 и 142
0,022
0,623
KNG1_QVVAGLNFR_vs_CRIS3_AVSPPAR
117 и 78
0,032
0,615
CATD_VSTLPAITLK_vs_CRIS3_AVSPPAR
58 и 78
0,020
0,625
KNG1_QVVAGLNFR_vs_CRIS3_YEDLYSNCK
117 и 79
0,016
0,630
CATD_VSTLPAITLK_vs_CRIS3_YEDLYSNCK
58 и 79
0,009
0,639
KNG1_QVVAGLNFR_vs_IBP3_YGQPLPGYTTK
117 и 100
0,019
0,626
CATD_VSTLPAITLK_vs_IBP3_FLNVLSPR
58 и 99
0,015
0,631
KNG1_QVVAGLNFR_vs_IGF2_GIVEECCFR
117 и 103
0,007
0,645
CATD_VSTLPAITLK_vs_IBP3_YGQPLPGYTTK
58 и 100
0,010
0,638
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,008
0,642
CATD_VSTLPAITLK_vs_IGF2_GIVEECCFR
58 и 103
0,005
0,652
KNG1_QVVAGLNFR_vs_PGRP2_AGLLRPDYALLGHR
117 и 126
0,045
0,608
CATD_VSTLPAITLK_vs_LYAM1_SYYWIGIR
58 и 120
0,026
0,620
LBP_ITGFLKPGK_vs_CRIS3_AVSPPAR
118 и 78
0,022
0,623
CATD_VSTLPAITLK_vs_PGRP2_AGLLRPDYALLGHR
58 и 126
0,021
0,624
LBP_ITGFLKPGK_vs_CRIS3_YEDLYSNCK
118 и 79
0,011
0,637
CATD_VSTLPAITLK_vs_SHBG_IALGGLLFPASNLR
58 и 18
0,023
0,622
LBP_ITGFLKPGK_vs_IBP3_YGQPLPGYTTK
118 и 100
0,032
0,615
CATD_VSTLPAITLK_vs_TENX_LNWEAPPGAFDSFLLR
58 и 141
0,007
0,646
LBP_ITGFLKPGK_vs_IGF2_GIVEECCFR
118 и 103
0,010
0,639
CATD_VSTLPAITLK_vs_TENX_LSQLSVTDVTTSSLR
58 и 142
0,017
0,628
LBP_ITGFLKPGK_vs_LYAM1_SYYWIGIR
118 и 120
0,016
0,630
CBPN_EALIQFLEQVHQGIK_vs_CRIS3_YEDLYSNCK
59 и 79
0,035
0,614
LBP_ITGFLKPGK_vs_PGRP2_AGLLRPDYALLGHR
118 и 126
0,025
0,620
CBPN_EALIQFLEQVHQGIK_vs_IBP3_YGQPLPGYTTK
59 и 100
0,035
0,613
LBP_ITGFLKPGK_vs_PSG3_VSAPSGTGHLPGLNPL
118 и 134
0,036
0,613
CBPN_EALIQFLEQVHQGIK_vs_IGF2_GIVEECCFR
59 и 103
0,029
0,617
LBP_ITGFLKPGK_vs_SHBG_IALGGLLFPASNLR
118 и 18
0,012
0,635
CBPN_EALIQFLEQVHQGIK_vs_LYAM1_SYYWIGIR
59 и 120
0,021
0,624
LBP_ITGFLKPGK_vs_SOM2.CSH_SVEGSCGF
118 и 139
0,026
0,619
CBPN_EALIQFLEQVHQGIK_vs_SHBG_IALGGLLFPASNLR
59 и 18
0,032
0,615
LBP_ITGFLKPGK_vs_TENX_LNWEAPPGAFDSFLLR
118 и 141
0,047
0,607
CBPN_EALIQFLEQVHQGIK_vs_TENX_LNWEAPPGAFDSFLLR
59 и 141
0,049
0,606
LBP_ITLPDFTGDLR_vs_ALS_IRPHTFTGLSGLR
119 и 40
0,047
0,607
CBPN_NNANGVDLNR_vs_CRIS3_YEDLYSNCK
60 и 79
0,027
0,619
LBP_ITLPDFTGDLR_vs_C163A_INPASLDK
119 и 54
0,031
0,616
CBPN_NNANGVDLNR_vs_IBP3_YGQPLPGYTTK
60 и 100
0,021
0,624
LBP_ITLPDFTGDLR_vs_CRIS3_AVSPPAR
119 и 78
0,006
0,648
CBPN_NNANGVDLNR_vs_IGF2_GIVEECCFR
60 и 103
0,008
0,642
LBP_ITLPDFTGDLR_vs_CRIS3_YEDLYSNCK
119 и 79
0,003
0,660
CBPN_NNANGVDLNR_vs_LYAM1_SYYWIGIR
60 и 120
0,019
0,626
LBP_ITLPDFTGDLR_vs_IBP2_LIQGAPTIR
119 и 98
0,048
0,606
CBPN_NNANGVDLNR_vs_SHBG_IALGGLLFPASNLR
60 и 18
0,033
0,615
LBP_ITLPDFTGDLR_vs_IBP3_FLNVLSPR
119 и 99
0,018
0,628
CD14_LTVGAAQVPAQLLVGALR_vs_CRIS3_YEDLYSNCK
61 и 79
0,028
0,618
LBP_ITLPDFTGDLR_vs_IBP3_YGQPLPGYTTK
119 и 100
0,007
0,645
CD14_LTVGAAQVPAQLLVGALR_vs_IBP3_FLNVLSPR
61 и 99
0,037
0,612
LBP_ITLPDFTGDLR_vs_IGF2_GIVEECCFR
119 и 103
0,003
0,658
CD14_LTVGAAQVPAQLLVGALR_vs_IBP3_YGQPLPGYTTK
61 и 100
0,028
0,618
LBP_ITLPDFTGDLR_vs_ITIH4_ILDDLSPR
119 и 112
0,022
0,623
CD14_LTVGAAQVPAQLLVGALR_vs_IGF2_GIVEECCFR
61 и 103
0,007
0,644
LBP_ITLPDFTGDLR_vs_LYAM1_SYYWIGIR
119 и 120
0,003
0,657
CD14_LTVGAAQVPAQLLVGALR_vs_LYAM1_SYYWIGIR
61 и 120
0,018
0,627
LBP_ITLPDFTGDLR_vs_NCAM1_GLGEISAASEFK
119 и 121
0,049
0,606
CD14_LTVGAAQVPAQLLVGALR_vs_PGRP2_AGLLRPDYALLGHR
61 и 126
0,050
0,606
LBP_ITLPDFTGDLR_vs_PGRP2_AGLLRPDYALLGHR
119 и 126
0,005
0,651
CD14_LTVGAAQVPAQLLVGALR_vs_SHBG_IALGGLLFPASNLR
61 и 18
0,047
0,607
LBP_ITLPDFTGDLR_vs_PSG3_VSAPSGTGHLPGLNPL
119 и 134
0,015
0,631
CD14_LTVGAAQVPAQLLVGALR_vs_TENX_LNWEAPPGAFDSFLLR
61 и 141
0,032
0,615
LBP_ITLPDFTGDLR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
119 и 135
0,039
0,611
CD14_SWLAELQQWLKPGLK_vs_IGF2_GIVEECCFR
62 и 103
0,027
0,619
LBP_ITLPDFTGDLR_vs_SHBG_IALGGLLFPASNLR
119 и 18
0,004
0,654
CD14_SWLAELQQWLKPGLK_vs_LYAM1_SYYWIGIR
62 и 120
0,030
0,617
LBP_ITLPDFTGDLR_vs_SOM2.CSH_SVEGSCGF
119 и 139
0,010
0,639
CFAB_YGLVTYATYPK_vs_CRIS3_YEDLYSNCK
64 и 79
0,033
0,615
LBP_ITLPDFTGDLR_vs_SPRL1_VLTHSELAPLR
119 и 140
0,030
0,617
CFAB_YGLVTYATYPK_vs_IBP3_FLNVLSPR
64 и 99
0,043
0,609
LBP_ITLPDFTGDLR_vs_TENX_LNWEAPPGAFDSFLLR
119 и 141
0,011
0,637
CFAB_YGLVTYATYPK_vs_IBP3_YGQPLPGYTTK
64 и 100
0,013
0,633
LBP_ITLPDFTGDLR_vs_TENX_LSQLSVTDVTTSSLR
119 и 142
0,021
0,624
CFAB_YGLVTYATYPK_vs_IGF2_GIVEECCFR
64 и 103
0,005
0,650
LBP_ITLPDFTGDLR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
119 и 144
0,049
0,606
CFAB_YGLVTYATYPK_vs_LYAM1_SYYWIGIR
64 и 120
0,020
0,625
LBP_ITLPDFTGDLR_vs_VTDB_ELPEHTVK
119 и 147
0,022
0,623
CFAB_YGLVTYATYPK_vs_SHBG_IALGGLLFPASNLR
64 и 18
0,041
0,610
PEDF_LQSLFDSPDFSK_vs_CRIS3_AVSPPAR
124 и 78
0,037
0,612
CLUS_ASSIIDELFQDR_vs_CRIS3_YEDLYSNCK
67 и 79
0,030
0,617
PEDF_LQSLFDSPDFSK_vs_CRIS3_YEDLYSNCK
124 и 79
0,019
0,626
CLUS_ASSIIDELFQDR_vs_IBP3_YGQPLPGYTTK
67 и 100
0,039
0,611
PEDF_LQSLFDSPDFSK_vs_IGF2_GIVEECCFR
124 и 103
0,037
0,612
CLUS_ASSIIDELFQDR_vs_IGF2_GIVEECCFR
67 и 103
0,017
0,629
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,025
0,620
CLUS_LFDSDPITVTVPVEVSR_vs_CRIS3_YEDLYSNCK
68 и 79
0,044
0,608
PEDF_LQSLFDSPDFSK_vs_TENX_LNWEAPPGAFDSFLLR
124 и 141
0,033
0,615
CLUS_LFDSDPITVTVPVEVSR_vs_IGF2_GIVEECCFR
68 и 103
0,046
0,607
PEDF_TVQAVLTVPK_vs_CRIS3_AVSPPAR
125 и 78
0,048
0,606
CO5_TLLPVSKPEIR_vs_CRIS3_AVSPPAR
70 и 78
0,047
0,607
PEDF_TVQAVLTVPK_vs_CRIS3_YEDLYSNCK
125 и 79
0,021
0,624
CO5_TLLPVSKPEIR_vs_CRIS3_YEDLYSNCK
70 и 79
0,023
0,622
PEDF_TVQAVLTVPK_vs_IBP3_YGQPLPGYTTK
125 и 100
0,049
0,606
CO5_TLLPVSKPEIR_vs_IBP3_YGQPLPGYTTK
70 и 100
0,043
0,609
PEDF_TVQAVLTVPK_vs_IGF2_GIVEECCFR
125 и 103
0,025
0,620
CO5_TLLPVSKPEIR_vs_IGF2_GIVEECCFR
70 и 103
0,014
0,632
PEDF_TVQAVLTVPK_vs_LYAM1_SYYWIGIR
125 и 120
0,038
0,611
CO5_TLLPVSKPEIR_vs_LYAM1_SYYWIGIR
70 и 120
0,019
0,626
PEDF_TVQAVLTVPK_vs_TENX_LNWEAPPGAFDSFLLR
125 и 141
0,049
0,606
CO5_TLLPVSKPEIR_vs_PGRP2_AGLLRPDYALLGHR
70 и 126
0,042
0,609
PRDX2_GLFIIDGK_vs_CRIS3_YEDLYSNCK
128 и 79
0,049
0,606
CO5_TLLPVSKPEIR_vs_SHBG_IALGGLLFPASNLR
70 и 18
0,032
0,615
PTGDS_GPGEDFR_vs_CRIS3_AVSPPAR
137 и 78
0,015
0,631
CO5_TLLPVSKPEIR_vs_TENX_LNWEAPPGAFDSFLLR
70 и 141
0,046
0,607
PTGDS_GPGEDFR_vs_CRIS3_YEDLYSNCK
137 и 79
0,007
0,646
CO5_VFQFLEK_vs_CRIS3_YEDLYSNCK
71 и 79
0,036
0,613
PTGDS_GPGEDFR_vs_IBP3_FLNVLSPR
137 и 99
0,042
0,609
CO5_VFQFLEK_vs_IGF2_GIVEECCFR
71 и 103
0,030
0,617
PTGDS_GPGEDFR_vs_IBP3_YGQPLPGYTTK
137 и 100
0,015
0,631
CO5_VFQFLEK_vs_LYAM1_SYYWIGIR
71 и 120
0,022
0,623
PTGDS_GPGEDFR_vs_IGF2_GIVEECCFR
137 и 103
0,004
0,655
CO5_VFQFLEK_vs_SHBG_IALGGLLFPASNLR
71 и 18
0,047
0,607
PTGDS_GPGEDFR_vs_LYAM1_SYYWIGIR
137 и 120
0,019
0,626
CO6_ALNHLPLEYNSALYSR_vs_IGF2_GIVEECCFR
72 и 103
0,037
0,612
PTGDS_GPGEDFR_vs_PGRP2_AGLLRPDYALLGHR
137 и 126
0,033
0,615
CO8A_SLLQPNK_vs_ALS_IRPHTFTGLSGLR
74 и 40
0,043
0,609
PTGDS_GPGEDFR_vs_SHBG_IALGGLLFPASNLR
137 и 18
0,021
0,624
CO8A_SLLQPNK_vs_CRIS3_YEDLYSNCK
74 и 79
0,015
0,631
PTGDS_GPGEDFR_vs_TENX_LNWEAPPGAFDSFLLR
137 и 141
0,012
0,636
CO8A_SLLQPNK_vs_IBP3_FLNVLSPR
74 и 99
0,024
0,622
PTGDS_GPGEDFR_vs_TENX_LSQLSVTDVTTSSLR
137 и 142
0,021
0,624
CO8A_SLLQPNK_vs_IBP3_YGQPLPGYTTK
74 и 100
0,009
0,640
THBG_AVLHIGEK_vs_CRIS3_YEDLYSNCK
143 и 79
0,028
0,618
CO8A_SLLQPNK_vs_IGF2_GIVEECCFR
74 и 103
0,002
0,663
THBG_AVLHIGEK_vs_IGF2_GIVEECCFR
143 и 103
0,026
0,620
CO8A_SLLQPNK_vs_LYAM1_SYYWIGIR
74 и 120
0,021
0,624
THBG_AVLHIGEK_vs_LYAM1_SYYWIGIR
143 и 120
0,025
0,620
CO8A_SLLQPNK_vs_PGRP2_AGLLRPDYALLGHR
74 и 126
0,036
0,613
THBG_AVLHIGEK_vs_SHBG_IALGGLLFPASNLR
143 и 18
0,047
0,607
CO8A_SLLQPNK_vs_SHBG_IALGGLLFPASNLR
74 и 18
0,028
0,618
VTNC_GQYCYELDEK_vs_CRIS3_AVSPPAR
149 и 78
0,032
0,616
CO8A_SLLQPNK_vs_TENX_LNWEAPPGAFDSFLLR
74 и 141
0,015
0,630
VTNC_GQYCYELDEK_vs_CRIS3_YEDLYSNCK
149 и 79
0,016
0,630
CO8A_SLLQPNK_vs_TENX_LSQLSVTDVTTSSLR
74 и 142
0,020
0,625
VTNC_GQYCYELDEK_vs_IBP3_FLNVLSPR
149 и 99
0,032
0,616
CO8B_QALEEFQK_vs_ALS_IRPHTFTGLSGLR
76 и 40
0,014
0,633
VTNC_GQYCYELDEK_vs_IBP3_YGQPLPGYTTK
149 и 100
0,006
0,647
CO8B_QALEEFQK_vs_CRIS3_AVSPPAR
76 и 78
0,028
0,618
VTNC_GQYCYELDEK_vs_IGF2_GIVEECCFR
149 и 103
0,004
0,656
CO8B_QALEEFQK_vs_CRIS3_YEDLYSNCK
76 и 79
0,013
0,634
VTNC_GQYCYELDEK_vs_ITIH4_ILDDLSPR
149 и 112
0,030
0,616
CO8B_QALEEFQK_vs_IBP3_FLNVLSPR
76 и 99
0,012
0,635
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,008
0,643
CO8B_QALEEFQK_vs_IBP3_YGQPLPGYTTK
76 и 100
0,007
0,645
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,033
0,614
CO8B_QALEEFQK_vs_IGF2_GIVEECCFR
76 и 103
0,001
0,676
VTNC_GQYCYELDEK_vs_PSG3_VSAPSGTGHLPGLNPL
149 и 134
0,036
0,613
CO8B_QALEEFQK_vs_LYAM1_SYYWIGIR
76 и 120
0,012
0,635
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,007
0,645
CO8B_QALEEFQK_vs_PGRP2_AGLLRPDYALLGHR
76 и 126
0,025
0,620
VTNC_GQYCYELDEK_vs_TENX_LNWEAPPGAFDSFLLR
149 и 141
0,032
0,615
CO8B_QALEEFQK_vs_PSG3_VSAPSGTGHLPGLNPL
76 и 134
0,027
0,619
VTNC_GQYCYELDEK_vs_VTDB_ELPEHTVK
149 и 147
0,043
0,609
CO8B_QALEEFQK_vs_SHBG_IALGGLLFPASNLR
76 и 18
0,027
0,619
VTNC_VDTVDPPYPR_vs_CRIS3_AVSPPAR
150 и 78
0,015
0,630
CO8B_QALEEFQK_vs_SPRL1_VLTHSELAPLR
76 и 140
0,041
0,610
VTNC_VDTVDPPYPR_vs_CRIS3_YEDLYSNCK
150 и 79
0,009
0,640
CO8B_QALEEFQK_vs_TENX_LNWEAPPGAFDSFLLR
76 и 141
0,008
0,643
VTNC_VDTVDPPYPR_vs_IBP3_FLNVLSPR
150 и 99
0,019
0,626
CO8B_QALEEFQK_vs_TENX_LSQLSVTDVTTSSLR
76 и 142
0,007
0,645
VTNC_VDTVDPPYPR_vs_IBP3_YGQPLPGYTTK
150 и 100
0,005
0,652
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_ALS_IRPHTFTGLSGLR
82 и 40
0,037
0,612
VTNC_VDTVDPPYPR_vs_IGF2_GIVEECCFR
150 и 103
0,004
0,655
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_C163A_INPASLDK
82 и 54
0,043
0,609
VTNC_VDTVDPPYPR_vs_ITIH4_ILDDLSPR
150 и 112
0,037
0,612
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_CRIS3_AVSPPAR
82 и 78
0,031
0,616
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,005
0,651
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_CRIS3_YEDLYSNCK
82 и 79
0,021
0,624
VTNC_VDTVDPPYPR_vs_PGRP2_AGLLRPDYALLGHR
150 и 126
0,022
0,623
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_IBP3_FLNVLSPR
82 и 99
0,021
0,624
VTNC_VDTVDPPYPR_vs_PSG3_VSAPSGTGHLPGLNPL
150 и 134
0,039
0,611
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_IBP3_YGQPLPGYTTK
82 и 100
0,013
0,634
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,005
0,650
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_IGF2_GIVEECCFR
82 и 103
0,007
0,644
VTNC_VDTVDPPYPR_vs_TENX_LNWEAPPGAFDSFLLR
150 и 141
0,026
0,620
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_ITIH4_ILDDLSPR
82 и 112
0,028
0,618
VTNC_VDTVDPPYPR_vs_VTDB_ELPEHTVK
150 и 147
0,030
0,616
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_LYAM1_SYYWIGIR
82 и 120
0,006
0,648
Таблица 57: эффективность классификации по обращениям, недели 19, 20 и 21
AUROC обращения для недель беременности от 19 0/7 до 21 6/7 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, без BMI-стратификации.
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
A2GL_DLLLPQPDLR_vs_LYAM1_SYYWIGIR
34 и 120
0,043
0,639
CD14_LTVGAAQVPAQLLVGALR_vs_VTDB_ELPEHTVK
61 и 147
0,009
0,680
A2GL_DLLLPQPDLR_vs_SHBG_IALGGLLFPASNLR
34 и 18
0,021
0,659
CD14_SWLAELQQWLKPGLK_vs_IBP1_VVESLAK
62 и 97
0,029
0,650
AFAM_DADPDTFFAK_vs_LYAM1_SYYWIGIR
37 и 120
0,018
0,662
CD14_SWLAELQQWLKPGLK_vs_LYAM1_SYYWIGIR
62 и 120
0,002
0,709
AFAM_DADPDTFFAK_vs_PGRP2_AGLLRPDYALLGHR
37 и 126
0,018
0,663
CD14_SWLAELQQWLKPGLK_vs_PGRP2_AGLLRPDYALLGHR
62 и 126
0,005
0,692
AFAM_DADPDTFFAK_vs_SHBG_IALGGLLFPASNLR
37 и 18
0,018
0,663
CD14_SWLAELQQWLKPGLK_vs_SHBG_IALGGLLFPASNLR
62 и 18
0,005
0,695
AFAM_HFQNLGK_vs_IBP1_VVESLAK
38 и 97
0,036
0,648
CD14_SWLAELQQWLKPGLK_vs_SPRL1_VLTHSELAPLR
62 и 140
0,025
0,654
AFAM_HFQNLGK_vs_LYAM1_SYYWIGIR
38 и 120
0,010
0,677
CD14_SWLAELQQWLKPGLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
62 и 144
0,028
0,652
AFAM_HFQNLGK_vs_PGRP2_AGLLRPDYALLGHR
38 и 126
0,017
0,664
CD14_SWLAELQQWLKPGLK_vs_VTDB_ELPEHTVK
62 и 147
0,007
0,684
AFAM_HFQNLGK_vs_SHBG_IALGGLLFPASNLR
38 и 18
0,014
0,670
CLUS_ASSIIDELFQDR_vs_IBP1_VVESLAK
67 и 97
0,048
0,636
ANGT_DPTFIPAPIQAK_vs_LYAM1_SYYWIGIR
42 и 120
0,030
0,649
CLUS_ASSIIDELFQDR_vs_LYAM1_SYYWIGIR
67 и 120
0,007
0,686
ANGT_DPTFIPAPIQAK_vs_SHBG_IALGGLLFPASNLR
42 и 18
0,013
0,671
CLUS_ASSIIDELFQDR_vs_PGRP2_AGLLRPDYALLGHR
67 и 126
0,016
0,665
APOC3_GWVTDGFSSLK_vs_ALS_IRPHTFTGLSGLR
47 и 40
0,040
0,641
CLUS_ASSIIDELFQDR_vs_SHBG_IALGGLLFPASNLR
67 и 18
0,006
0,691
APOC3_GWVTDGFSSLK_vs_CRIS3_AVSPPAR
47 и 78
0,038
0,643
CLUS_ASSIIDELFQDR_vs_VTDB_ELPEHTVK
67 и 147
0,036
0,644
APOC3_GWVTDGFSSLK_vs_CRIS3_YEDLYSNCK
47 и 79
0,038
0,643
CLUS_LFDSDPITVTVPVEVSR_vs_IBP1_VVESLAK
68 и 97
0,047
0,637
APOC3_GWVTDGFSSLK_vs_CSH_AHQLAIDTYQEFEETYIPK
47 и 80
0,040
0,641
CLUS_LFDSDPITVTVPVEVSR_vs_LYAM1_SYYWIGIR
68 и 120
0,006
0,688
APOC3_GWVTDGFSSLK_vs_IBP1_VVESLAK
47 и 97
0,023
0,656
CLUS_LFDSDPITVTVPVEVSR_vs_PGRP2_AGLLRPDYALLGHR
68 и 126
0,023
0,656
APOC3_GWVTDGFSSLK_vs_IBP3_YGQPLPGYTTK
47 и 100
0,037
0,644
CLUS_LFDSDPITVTVPVEVSR_vs_SHBG_IALGGLLFPASNLR
68 и 18
0,007
0,687
APOC3_GWVTDGFSSLK_vs_IGF2_GIVEECCFR
47 и 103
0,048
0,636
CLUS_LFDSDPITVTVPVEVSR_vs_VTDB_ELPEHTVK
68 и 147
0,020
0,660
APOC3_GWVTDGFSSLK_vs_ITIH4_ILDDLSPR
47 и 112
0,032
0,647
CO5_TLLPVSKPEIR_vs_LYAM1_SYYWIGIR
70 и 120
0,012
0,674
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,005
0,693
CO5_TLLPVSKPEIR_vs_PGRP2_AGLLRPDYALLGHR
70 и 126
0,043
0,639
APOC3_GWVTDGFSSLK_vs_PGRP2_AGLLRPDYALLGHR
47 и 126
0,011
0,675
CO5_TLLPVSKPEIR_vs_SHBG_IALGGLLFPASNLR
70 и 18
0,016
0,666
APOC3_GWVTDGFSSLK_vs_SHBG_IALGGLLFPASNLR
47 и 18
0,011
0,674
CO5_TLLPVSKPEIR_vs_SPRL1_VLTHSELAPLR
70 и 140
0,042
0,640
APOC3_GWVTDGFSSLK_vs_SOM2.CSH_SVEGSCGF
47 и 139
0,038
0,643
CO5_VFQFLEK_vs_LYAM1_SYYWIGIR
71 и 120
0,025
0,654
APOC3_GWVTDGFSSLK_vs_SPRL1_VLTHSELAPLR
47 и 140
0,027
0,653
CO5_VFQFLEK_vs_SHBG_IALGGLLFPASNLR
71 и 18
0,023
0,656
APOC3_GWVTDGFSSLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
47 и 144
0,027
0,652
CO6_ALNHLPLEYNSALYSR_vs_LYAM1_SYYWIGIR
72 и 120
0,043
0,639
APOC3_GWVTDGFSSLK_vs_VTDB_ELPEHTVK
47 и 147
0,026
0,653
CO6_ALNHLPLEYNSALYSR_vs_SHBG_IALGGLLFPASNLR
72 и 18
0,027
0,653
APOH_ATVVYQGER_vs_CRIS3_YEDLYSNCK
48 и 79
0,035
0,645
CO8A_SLLQPNK_vs_LYAM1_SYYWIGIR
74 и 120
0,013
0,670
APOH_ATVVYQGER_vs_IBP1_VVESLAK
48 и 97
0,021
0,659
CO8A_SLLQPNK_vs_PGRP2_AGLLRPDYALLGHR
74 и 126
0,026
0,653
APOH_ATVVYQGER_vs_ITIH4_ILDDLSPR
48 и 112
0,025
0,655
CO8A_SLLQPNK_vs_SHBG_IALGGLLFPASNLR
74 и 18
0,022
0,658
APOH_ATVVYQGER_vs_LYAM1_SYYWIGIR
48 и 120
0,002
0,712
CO8B_QALEEFQK_vs_IBP1_VVESLAK
76 и 97
0,044
0,639
APOH_ATVVYQGER_vs_PGRP2_AGLLRPDYALLGHR
48 и 126
0,002
0,717
CO8B_QALEEFQK_vs_LYAM1_SYYWIGIR
76 и 120
0,012
0,673
APOH_ATVVYQGER_vs_PSG3_VSAPSGTGHLPGLNPL
48 и 134
0,020
0,661
CO8B_QALEEFQK_vs_PGRP2_AGLLRPDYALLGHR
76 и 126
0,030
0,649
APOH_ATVVYQGER_vs_SHBG_IALGGLLFPASNLR
48 и 18
0,003
0,704
CO8B_QALEEFQK_vs_SHBG_IALGGLLFPASNLR
76 и 18
0,024
0,655
APOH_ATVVYQGER_vs_SPRL1_VLTHSELAPLR
48 и 140
0,011
0,674
CO8B_QALEEFQK_vs_SPRL1_VLTHSELAPLR
76 и 140
0,040
0,641
APOH_ATVVYQGER_vs_VTDB_ELPEHTVK
48 и 147
0,007
0,684
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_LYAM1_SYYWIGIR
82 и 120
0,036
0,644
B2MG_VEHSDLSFSK_vs_LYAM1_SYYWIGIR
50 и 120
0,007
0,687
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_PGRP2_AGLLRPDYALLGHR
82 и 126
0,037
0,643
B2MG_VEHSDLSFSK_vs_PGRP2_AGLLRPDYALLGHR
50 и 126
0,044
0,639
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_SHBG_IALGGLLFPASNLR
82 и 18
0,029
0,650
B2MG_VEHSDLSFSK_vs_SHBG_IALGGLLFPASNLR
50 и 18
0,012
0,673
ENPP2_TYLHTYESEI_vs_LYAM1_SYYWIGIR
83 и 120
0,021
0,659
B2MG_VNHVTLSQPK_vs_IBP1_VVESLAK
51 и 97
0,046
0,638
ENPP2_TYLHTYESEI_vs_PGRP2_AGLLRPDYALLGHR
83 и 126
0,025
0,655
B2MG_VNHVTLSQPK_vs_LYAM1_SYYWIGIR
51 и 120
0,002
0,717
ENPP2_TYLHTYESEI_vs_SHBG_IALGGLLFPASNLR
83 и 18
0,026
0,653
B2MG_VNHVTLSQPK_vs_PGRP2_AGLLRPDYALLGHR
51 и 126
0,009
0,681
F13B_GDTYPAELYITGSILR_vs_CRIS3_AVSPPAR
84 и 78
0,044
0,639
B2MG_VNHVTLSQPK_vs_SHBG_IALGGLLFPASNLR
51 и 18
0,005
0,692
F13B_GDTYPAELYITGSILR_vs_CRIS3_YEDLYSNCK
84 и 79
0,039
0,643
B2MG_VNHVTLSQPK_vs_SPRL1_VLTHSELAPLR
51 и 140
0,023
0,656
F13B_GDTYPAELYITGSILR_vs_IBP1_VVESLAK
84 и 97
0,033
0,647
BGH3_LTLLAPLNSVFK_vs_LYAM1_SYYWIGIR
52 и 120
0,039
0,642
F13B_GDTYPAELYITGSILR_vs_LYAM1_SYYWIGIR
84 и 120
0,001
0,732
BGH3_LTLLAPLNSVFK_vs_SHBG_IALGGLLFPASNLR
52 и 18
0,049
0,635
F13B_GDTYPAELYITGSILR_vs_PGRP2_AGLLRPDYALLGHR
84 и 126
0,005
0,694
CAH1_GGPFSDSYR_vs_IBP1_VVESLAK
56 и 97
0,033
0,647
F13B_GDTYPAELYITGSILR_vs_SHBG_IALGGLLFPASNLR
84 и 18
0,003
0,708
CAH1_GGPFSDSYR_vs_LYAM1_SYYWIGIR
56 и 120
0,015
0,668
F13B_GDTYPAELYITGSILR_vs_SPRL1_VLTHSELAPLR
84 и 140
0,007
0,685
CAH1_GGPFSDSYR_vs_PGRP2_AGLLRPDYALLGHR
56 и 126
0,020
0,661
F13B_GDTYPAELYITGSILR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
84 и 144
0,046
0,638
CAH1_GGPFSDSYR_vs_SHBG_IALGGLLFPASNLR
56 и 18
0,008
0,682
F13B_GDTYPAELYITGSILR_vs_VTDB_ELPEHTVK
84 и 147
0,037
0,644
CATD_VGFAEAAR_vs_ALS_IRPHTFTGLSGLR
57 и 40
0,001
0,720
FETUA_FSVVYAK_vs_LYAM1_SYYWIGIR
88 и 120
0,012
0,674
CATD_VGFAEAAR_vs_C163A_INPASLDK
57 и 54
0,000
0,772
FETUA_FSVVYAK_vs_SHBG_IALGGLLFPASNLR
88 и 18
0,017
0,664
CATD_VGFAEAAR_vs_CHL1_VIAVNEVGR
57 и 66
0,045
0,638
FETUA_HTLNQIDEVK_vs_LYAM1_SYYWIGIR
89 и 120
0,023
0,657
CATD_VGFAEAAR_vs_CRIS3_AVSPPAR
57 и 78
0,004
0,700
FETUA_HTLNQIDEVK_vs_SHBG_IALGGLLFPASNLR
89 и 18
0,019
0,662
CATD_VGFAEAAR_vs_CRIS3_YEDLYSNCK
57 и 79
0,004
0,700
HEMO_NFPSPVDAAFR_vs_LYAM1_SYYWIGIR
93 и 120
0,014
0,668
CATD_VGFAEAAR_vs_CSH_AHQLAIDTYQEFEETYIPK
57 и 80
0,001
0,739
HEMO_NFPSPVDAAFR_vs_PGRP2_AGLLRPDYALLGHR
93 и 126
0,046
0,637
CATD_VGFAEAAR_vs_CSH_ISLLLIESWLEPVR
57 и 81
0,001
0,733
HEMO_NFPSPVDAAFR_vs_SHBG_IALGGLLFPASNLR
93 и 18
0,008
0,683
CATD_VGFAEAAR_vs_FBLN1_TGYYFDGISR
57 и 86
0,001
0,728
IBP4_QCHPALDGQR_vs_CRIS3_AVSPPAR
2 и 78
0,024
0,655
CATD_VGFAEAAR_vs_IBP1_VVESLAK
57 и 97
0,003
0,702
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,024
0,656
CATD_VGFAEAAR_vs_IBP2_LIQGAPTIR
57 и 98
0,010
0,678
IBP4_QCHPALDGQR_vs_IBP1_VVESLAK
2 и 97
0,026
0,653
CATD_VGFAEAAR_vs_IBP3_FLNVLSPR
57 и 99
0,005
0,694
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,002
0,713
CATD_VGFAEAAR_vs_IBP3_YGQPLPGYTTK
57 и 100
0,002
0,708
IBP4_QCHPALDGQR_vs_PGRP2_AGLLRPDYALLGHR
2 и 126
0,008
0,683
CATD_VGFAEAAR_vs_IGF2_GIVEECCFR
57 и 103
0,001
0,721
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,002
0,709
CATD_VGFAEAAR_vs_ITIH4_ILDDLSPR
57 и 112
0,001
0,739
IBP4_QCHPALDGQR_vs_SPRL1_VLTHSELAPLR
2 и 140
0,015
0,667
CATD_VGFAEAAR_vs_LYAM1_SYYWIGIR
57 и 120
0,000
0,778
IBP6_GAQTLYVPNCDHR_vs_PGRP2_AGLLRPDYALLGHR
101 и 126
0,030
0,650
CATD_VGFAEAAR_vs_NCAM1_GLGEISAASEFK
57 и 121
0,004
0,701
IBP6_GAQTLYVPNCDHR_vs_SHBG_IALGGLLFPASNLR
101 и 18
0,013
0,671
CATD_VGFAEAAR_vs_PGRP2_AGLLRPDYALLGHR
57 и 126
0,000
0,773
IBP6_HLDSVLQQLQTEVYR_vs_LYAM1_SYYWIGIR
102 и 120
0,020
0,660
CATD_VGFAEAAR_vs_PRG2_WNFAYWAAHQPWSR
57 и 129
0,010
0,677
IBP6_HLDSVLQQLQTEVYR_vs_SHBG_IALGGLLFPASNLR
102 и 18
0,010
0,677
CATD_VGFAEAAR_vs_PSG1_FQLPGQK
57 и 131
0,001
0,719
INHBC_LDFHFSSDR_vs_LYAM1_SYYWIGIR
107 и 120
0,046
0,637
CATD_VGFAEAAR_vs_PSG3_VSAPSGTGHLPGLNPL
57 и 134
0,001
0,729
INHBC_LDFHFSSDR_vs_PGRP2_AGLLRPDYALLGHR
107 и 126
0,028
0,652
CATD_VGFAEAAR_vs_SHBG_IALGGLLFPASNLR
57 и 18
0,000
0,749
INHBC_LDFHFSSDR_vs_SHBG_IALGGLLFPASNLR
107 и 18
0,033
0,647
CATD_VGFAEAAR_vs_SOM2.CSH_NYGLLYCFR
57 и 138
0,001
0,723
ITIH3_ALDLSLK_vs_SHBG_IALGGLLFPASNLR
111 и 18
0,026
0,653
CATD_VGFAEAAR_vs_SOM2.CSH_SVEGSCGF
57 и 139
0,001
0,722
KNG1_DIPTNSPELEETLTHTITK_vs_LYAM1_SYYWIGIR
116 и 120
0,017
0,665
CATD_VGFAEAAR_vs_SPRL1_VLTHSELAPLR
57 и 140
0,000
0,762
KNG1_DIPTNSPELEETLTHTITK_vs_SHBG_IALGGLLFPASNLR
116 и 18
0,027
0,653
CATD_VGFAEAAR_vs_TENX_LNWEAPPGAFDSFLLR
57 и 141
0,000
0,757
KNG1_QVVAGLNFR_vs_IBP1_VVESLAK
117 и 97
0,032
0,648
CATD_VGFAEAAR_vs_TENX_LSQLSVTDVTTSSLR
57 и 142
0,000
0,744
KNG1_QVVAGLNFR_vs_ITIH4_ILDDLSPR
117 и 112
0,028
0,651
CATD_VGFAEAAR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
57 и 144
0,000
0,743
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,002
0,711
CATD_VGFAEAAR_vs_VTDB_ELPEHTVK
57 и 147
0,001
0,739
KNG1_QVVAGLNFR_vs_PGRP2_AGLLRPDYALLGHR
117 и 126
0,010
0,677
CATD_VSTLPAITLK_vs_ALS_IRPHTFTGLSGLR
58 и 40
0,001
0,728
KNG1_QVVAGLNFR_vs_SHBG_IALGGLLFPASNLR
117 и 18
0,003
0,703
CATD_VSTLPAITLK_vs_C163A_INPASLDK
58 и 54
0,001
0,725
KNG1_QVVAGLNFR_vs_SPRL1_VLTHSELAPLR
117 и 140
0,019
0,662
CATD_VSTLPAITLK_vs_CRIS3_AVSPPAR
58 и 78
0,004
0,698
PEDF_LQSLFDSPDFSK_vs_IBP1_VVESLAK
124 и 97
0,044
0,639
CATD_VSTLPAITLK_vs_CRIS3_YEDLYSNCK
58 и 79
0,004
0,700
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,004
0,697
CATD_VSTLPAITLK_vs_CSH_AHQLAIDTYQEFEETYIPK
58 и 80
0,001
0,724
PEDF_LQSLFDSPDFSK_vs_PGRP2_AGLLRPDYALLGHR
124 и 126
0,043
0,640
CATD_VSTLPAITLK_vs_CSH_ISLLLIESWLEPVR
58 и 81
0,002
0,716
PEDF_LQSLFDSPDFSK_vs_SHBG_IALGGLLFPASNLR
124 и 18
0,017
0,665
CATD_VSTLPAITLK_vs_FBLN1_TGYYFDGISR
58 и 86
0,001
0,728
PEDF_TVQAVLTVPK_vs_LYAM1_SYYWIGIR
125 и 120
0,003
0,702
CATD_VSTLPAITLK_vs_IBP1_VVESLAK
58 и 97
0,004
0,700
PEDF_TVQAVLTVPK_vs_PGRP2_AGLLRPDYALLGHR
125 и 126
0,025
0,654
CATD_VSTLPAITLK_vs_IBP2_LIQGAPTIR
58 и 98
0,009
0,680
PEDF_TVQAVLTVPK_vs_SHBG_IALGGLLFPASNLR
125 и 18
0,012
0,673
CATD_VSTLPAITLK_vs_IBP3_FLNVLSPR
58 и 99
0,003
0,707
PEDF_TVQAVLTVPK_vs_SPRL1_VLTHSELAPLR
125 и 140
0,036
0,645
CATD_VSTLPAITLK_vs_IBP3_YGQPLPGYTTK
58 и 100
0,001
0,719
PRDX2_GLFIIDGK_vs_CRIS3_AVSPPAR
128 и 78
0,028
0,651
CATD_VSTLPAITLK_vs_IGF2_GIVEECCFR
58 и 103
0,001
0,732
PRDX2_GLFIIDGK_vs_CRIS3_YEDLYSNCK
128 и 79
0,026
0,653
CATD_VSTLPAITLK_vs_ITIH4_ILDDLSPR
58 и 112
0,001
0,724
PRDX2_GLFIIDGK_vs_IBP1_VVESLAK
128 и 97
0,024
0,655
CATD_VSTLPAITLK_vs_LYAM1_SYYWIGIR
58 и 120
0,000
0,771
PRDX2_GLFIIDGK_vs_IBP2_LIQGAPTIR
128 и 98
0,040
0,641
CATD_VSTLPAITLK_vs_NCAM1_GLGEISAASEFK
58 и 121
0,003
0,704
PRDX2_GLFIIDGK_vs_LYAM1_SYYWIGIR
128 и 120
0,009
0,681
CATD_VSTLPAITLK_vs_PGRP2_AGLLRPDYALLGHR
58 и 126
0,000
0,775
PRDX2_GLFIIDGK_vs_PGRP2_AGLLRPDYALLGHR
128 и 126
0,009
0,679
CATD_VSTLPAITLK_vs_PRG2_WNFAYWAAHQPWSR
58 и 129
0,028
0,652
PRDX2_GLFIIDGK_vs_SHBG_IALGGLLFPASNLR
128 и 18
0,004
0,697
CATD_VSTLPAITLK_vs_PSG1_FQLPGQK
58 и 131
0,005
0,693
PRDX2_GLFIIDGK_vs_SPRL1_VLTHSELAPLR
128 и 140
0,042
0,640
CATD_VSTLPAITLK_vs_PSG3_VSAPSGTGHLPGLNPL
58 и 134
0,001
0,737
PRDX2_GLFIIDGK_vs_TENX_LNWEAPPGAFDSFLLR
128 и 141
0,038
0,643
CATD_VSTLPAITLK_vs_SHBG_IALGGLLFPASNLR
58 и 18
0,000
0,755
PRDX2_GLFIIDGK_vs_VTDB_ELPEHTVK
128 и 147
0,041
0,641
CATD_VSTLPAITLK_vs_SOM2.CSH_NYGLLYCFR
58 и 138
0,003
0,707
PSG2_IHPSYTNYR_vs_SHBG_IALGGLLFPASNLR
133 и 18
0,049
0,635
CATD_VSTLPAITLK_vs_SOM2.CSH_SVEGSCGF
58 и 139
0,004
0,696
PTGDS_GPGEDFR_vs_IBP1_VVESLAK
137 и 97
0,039
0,642
CATD_VSTLPAITLK_vs_SPRL1_VLTHSELAPLR
58 и 140
0,000
0,753
PTGDS_GPGEDFR_vs_LYAM1_SYYWIGIR
137 и 120
0,009
0,681
CATD_VSTLPAITLK_vs_TENX_LNWEAPPGAFDSFLLR
58 и 141
0,000
0,765
PTGDS_GPGEDFR_vs_PGRP2_AGLLRPDYALLGHR
137 и 126
0,017
0,665
CATD_VSTLPAITLK_vs_TENX_LSQLSVTDVTTSSLR
58 и 142
0,000
0,742
PTGDS_GPGEDFR_vs_SHBG_IALGGLLFPASNLR
137 и 18
0,010
0,677
CATD_VSTLPAITLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
58 и 144
0,001
0,729
PTGDS_GPGEDFR_vs_SOM2.CSH_SVEGSCGF
137 и 139
0,048
0,636
CATD_VSTLPAITLK_vs_VTDB_ELPEHTVK
58 и 147
0,000
0,745
PTGDS_GPGEDFR_vs_SPRL1_VLTHSELAPLR
137 и 140
0,035
0,645
CBPN_EALIQFLEQVHQGIK_vs_LYAM1_SYYWIGIR
59 и 120
0,042
0,640
THBG_AVLHIGEK_vs_LYAM1_SYYWIGIR
143 и 120
0,018
0,663
CBPN_EALIQFLEQVHQGIK_vs_SHBG_IALGGLLFPASNLR
59 и 18
0,011
0,674
THBG_AVLHIGEK_vs_PGRP2_AGLLRPDYALLGHR
143 и 126
0,025
0,655
CBPN_NNANGVDLNR_vs_LYAM1_SYYWIGIR
60 и 120
0,020
0,660
THBG_AVLHIGEK_vs_SHBG_IALGGLLFPASNLR
143 и 18
0,023
0,657
CBPN_NNANGVDLNR_vs_SHBG_IALGGLLFPASNLR
60 и 18
0,019
0,661
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,008
0,684
CBPN_NNANGVDLNR_vs_SPRL1_VLTHSELAPLR
60 и 140
0,016
0,665
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,020
0,660
CD14_LTVGAAQVPAQLLVGALR_vs_IBP1_VVESLAK
61 и 97
0,028
0,651
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,012
0,674
CD14_LTVGAAQVPAQLLVGALR_vs_LYAM1_SYYWIGIR
61 и 120
0,002
0,717
VTNC_GQYCYELDEK_vs_SPRL1_VLTHSELAPLR
149 и 140
0,047
0,637
CD14_LTVGAAQVPAQLLVGALR_vs_PGRP2_AGLLRPDYALLGHR
61 и 126
0,004
0,699
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,008
0,684
CD14_LTVGAAQVPAQLLVGALR_vs_SHBG_IALGGLLFPASNLR
61 и 18
0,003
0,703
VTNC_VDTVDPPYPR_vs_PGRP2_AGLLRPDYALLGHR
150 и 126
0,019
0,661
CD14_LTVGAAQVPAQLLVGALR_vs_SOM2.CSH_SVEGSCGF
61 и 139
0,049
0,635
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,007
0,686
CD14_LTVGAAQVPAQLLVGALR_vs_SPRL1_VLTHSELAPLR
61 и 140
0,020
0,660
VTNC_VDTVDPPYPR_vs_VTDB_ELPEHTVK
150 и 147
0,030
0,649
CD14_LTVGAAQVPAQLLVGALR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
61 и 144
0,031
0,648
Таблица 58: эффективность классификации по обращениям, недели 19, 20 и 21
AUROC обращения для недель беременности от 19 0/7 до 21 6/7 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, с BMI-стратификацией (>22≤37).
Обращение
SEQ ID №
p-зн.
ROC_ AUC
Обращение
SEQ ID №
p-зн,
ROC_ AUC
A2GL_DLLLPQPDLR_vs_LYAM1_SYYWIGIR
34 и 120
0,025
0,688
CD14_SWLAELQQWLKPGLK_vs_SHBG_IALGGLLFPASNLR
62 и 18
0,010
0,716
A2GL_DLLLPQPDLR_vs_PGRP2_AGLLRPDYALLGHR
34 и 126
0,029
0,683
CLUS_ASSIIDELFQDR_vs_LYAM1_SYYWIGIR
67 и 120
0,020
0,695
A2GL_DLLLPQPDLR_vs_SHBG_IALGGLLFPASNLR
34 и 18
0,026
0,687
CLUS_ASSIIDELFQDR_vs_PGRP2_AGLLRPDYALLGHR
67 и 126
0,014
0,705
AFAM_DADPDTFFAK_vs_LYAM1_SYYWIGIR
37 и 120
0,011
0,714
CLUS_ASSIIDELFQDR_vs_SHBG_IALGGLLFPASNLR
67 и 18
0,011
0,712
AFAM_DADPDTFFAK_vs_PGRP2_AGLLRPDYALLGHR
37 и 126
0,002
0,755
CLUS_LFDSDPITVTVPVEVSR_vs_LYAM1_SYYWIGIR
68 и 120
0,044
0,668
AFAM_DADPDTFFAK_vs_SHBG_IALGGLLFPASNLR
37 и 18
0,006
0,730
CLUS_LFDSDPITVTVPVEVSR_vs_PGRP2_AGLLRPDYALLGHR
68 и 126
0,045
0,668
AFAM_DADPDTFFAK_vs_VTDB_ELPEHTVK
37 и 147
0,024
0,689
CLUS_LFDSDPITVTVPVEVSR_vs_SHBG_IALGGLLFPASNLR
68 и 18
0,020
0,694
AFAM_HFQNLGK_vs_IBP1_VVESLAK
38 и 97
0,034
0,684
CO5_TLLPVSKPEIR_vs_LYAM1_SYYWIGIR
70 и 120
0,039
0,672
AFAM_HFQNLGK_vs_ITIH4_ILDDLSPR
38 и 112
0,025
0,688
CO5_TLLPVSKPEIR_vs_SHBG_IALGGLLFPASNLR
70 и 18
0,018
0,698
AFAM_HFQNLGK_vs_LYAM1_SYYWIGIR
38 и 120
0,004
0,744
CO5_VFQFLEK_vs_SHBG_IALGGLLFPASNLR
71 и 18
0,039
0,672
AFAM_HFQNLGK_vs_PGRP2_AGLLRPDYALLGHR
38 и 126
0,001
0,770
CO6_ALNHLPLEYNSALYSR_vs_SHBG_IALGGLLFPASNLR
72 и 18
0,022
0,692
AFAM_HFQNLGK_vs_SHBG_IALGGLLFPASNLR
38 и 18
0,004
0,738
CO8A_SLLQPNK_vs_LYAM1_SYYWIGIR
74 и 120
0,044
0,669
AFAM_HFQNLGK_vs_SOM2.CSH_SVEGSCGF
38 и 139
0,023
0,690
CO8A_SLLQPNK_vs_PGRP2_AGLLRPDYALLGHR
74 и 126
0,032
0,679
AFAM_HFQNLGK_vs_VTDB_ELPEHTVK
38 и 147
0,009
0,719
CO8A_SLLQPNK_vs_SHBG_IALGGLLFPASNLR
74 и 18
0,032
0,679
ANGT_DPTFIPAPIQAK_vs_LYAM1_SYYWIGIR
42 и 120
0,023
0,690
CO8B_QALEEFQK_vs_LYAM1_SYYWIGIR
76 и 120
0,041
0,671
ANGT_DPTFIPAPIQAK_vs_PGRP2_AGLLRPDYALLGHR
42 и 126
0,037
0,675
CO8B_QALEEFQK_vs_PGRP2_AGLLRPDYALLGHR
76 и 126
0,037
0,674
ANGT_DPTFIPAPIQAK_vs_SHBG_IALGGLLFPASNLR
42 и 18
0,006
0,730
CO8B_QALEEFQK_vs_SHBG_IALGGLLFPASNLR
76 и 18
0,039
0,672
APOH_ATVVYQGER_vs_IBP1_VVESLAK
48 и 97
0,049
0,665
F13B_GDTYPAELYITGSILR_vs_CRIS3_YEDLYSNCK
84 и 79
0,050
0,664
APOH_ATVVYQGER_vs_ITIH4_ILDDLSPR
48 и 112
0,049
0,665
F13B_GDTYPAELYITGSILR_vs_LYAM1_SYYWIGIR
84 и 120
0,002
0,765
APOH_ATVVYQGER_vs_LYAM1_SYYWIGIR
48 и 120
0,006
0,729
F13B_GDTYPAELYITGSILR_vs_PGRP2_AGLLRPDYALLGHR
84 и 126
0,002
0,755
APOH_ATVVYQGER_vs_PGRP2_AGLLRPDYALLGHR
48 и 126
0,001
0,788
F13B_GDTYPAELYITGSILR_vs_SHBG_IALGGLLFPASNLR
84 и 18
0,002
0,764
APOH_ATVVYQGER_vs_SHBG_IALGGLLFPASNLR
48 и 18
0,003
0,750
F13B_GDTYPAELYITGSILR_vs_VTDB_ELPEHTVK
84 и 147
0,041
0,671
APOH_ATVVYQGER_vs_VTDB_ELPEHTVK
48 и 147
0,012
0,711
FETUA_FSVVYAK_vs_LYAM1_SYYWIGIR
88 и 120
0,039
0,673
B2MG_VEHSDLSFSK_vs_LYAM1_SYYWIGIR
50 и 120
0,027
0,686
FETUA_FSVVYAK_vs_SHBG_IALGGLLFPASNLR
88 и 18
0,019
0,697
B2MG_VEHSDLSFSK_vs_SHBG_IALGGLLFPASNLR
50 и 18
0,027
0,685
FETUA_HTLNQIDEVK_vs_LYAM1_SYYWIGIR
89 и 120
0,034
0,678
B2MG_VNHVTLSQPK_vs_LYAM1_SYYWIGIR
51 и 120
0,004
0,740
FETUA_HTLNQIDEVK_vs_SHBG_IALGGLLFPASNLR
89 и 18
0,018
0,699
B2MG_VNHVTLSQPK_vs_PGRP2_AGLLRPDYALLGHR
51 и 126
0,009
0,718
HEMO_NFPSPVDAAFR_vs_LYAM1_SYYWIGIR
93 и 120
0,042
0,670
B2MG_VNHVTLSQPK_vs_SHBG_IALGGLLFPASNLR
51 и 18
0,006
0,731
HEMO_NFPSPVDAAFR_vs_SHBG_IALGGLLFPASNLR
93 и 18
0,015
0,704
BGH3_LTLLAPLNSVFK_vs_LYAM1_SYYWIGIR
52 и 120
0,043
0,670
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,048
0,666
BGH3_LTLLAPLNSVFK_vs_PGRP2_AGLLRPDYALLGHR
52 и 126
0,027
0,685
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,002
0,756
BGH3_LTLLAPLNSVFK_vs_SHBG_IALGGLLFPASNLR
52 и 18
0,027
0,685
IBP4_QCHPALDGQR_vs_PGRP2_AGLLRPDYALLGHR
2 и 126
0,004
0,741
CAH1_GGPFSDSYR_vs_LYAM1_SYYWIGIR
56 и 120
0,041
0,671
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,002
0,760
CAH1_GGPFSDSYR_vs_PGRP2_AGLLRPDYALLGHR
56 и 126
0,041
0,671
IBP6_GAQTLYVPNCDHR_vs_PGRP2_AGLLRPDYALLGHR
101 и 126
0,019
0,697
CAH1_GGPFSDSYR_vs_SHBG_IALGGLLFPASNLR
56 и 18
0,013
0,708
IBP6_GAQTLYVPNCDHR_vs_SHBG_IALGGLLFPASNLR
101 и 18
0,024
0,689
CATD_VGFAEAAR_vs_ALS_IRPHTFTGLSGLR
57 и 40
0,008
0,723
IBP6_HLDSVLQQLQTEVYR_vs_LYAM1_SYYWIGIR
102 и 120
0,014
0,706
CATD_VGFAEAAR_vs_C163A_INPASLDK
57 и 54
0,000
0,791
IBP6_HLDSVLQQLQTEVYR_vs_PGRP2_AGLLRPDYALLGHR
102 и 126
0,027
0,686
CATD_VGFAEAAR_vs_CRIS3_AVSPPAR
57 и 78
0,010
0,716
IBP6_HLDSVLQQLQTEVYR_vs_SHBG_IALGGLLFPASNLR
102 и 18
0,009
0,720
CATD_VGFAEAAR_vs_CRIS3_YEDLYSNCK
57 и 79
0,009
0,717
INHBC_LDFHFSSDR_vs_PGRP2_AGLLRPDYALLGHR
107 и 126
0,037
0,675
CATD_VGFAEAAR_vs_CSH_AHQLAIDTYQEFEETYIPK
57 и 80
0,001
0,773
ITIH3_ALDLSLK_vs_SHBG_IALGGLLFPASNLR
111 и 18
0,021
0,693
CATD_VGFAEAAR_vs_CSH_ISLLLIESWLEPVR
57 и 81
0,001
0,771
ITIH4_NPLVWVHASPEHVVVTR_vs_ALS_IRPHTFTGLSGLR
113 и 40
0,034
0,678
CATD_VGFAEAAR_vs_FBLN1_TGYYFDGISR
57 и 86
0,034
0,677
ITIH4_NPLVWVHASPEHVVVTR_vs_CHL1_VIAVNEVGR
113 и 66
0,045
0,667
CATD_VGFAEAAR_vs_IBP1_VVESLAK
57 и 97
0,012
0,711
ITIH4_NPLVWVHASPEHVVVTR_vs_CSH_AHQLAIDTYQEFEETYIPK
113 и 80
0,044
0,668
CATD_VGFAEAAR_vs_IBP2_LIQGAPTIR
57 и 98
0,016
0,701
ITIH4_NPLVWVHASPEHVVVTR_vs_FBLN1_TGYYFDGISR
113 и 86
0,016
0,701
CATD_VGFAEAAR_vs_IBP3_FLNVLSPR
57 и 99
0,018
0,698
ITIH4_NPLVWVHASPEHVVVTR_vs_IBP3_FLNVLSPR
113 и 99
0,025
0,688
CATD_VGFAEAAR_vs_IBP3_YGQPLPGYTTK
57 и 100
0,014
0,705
ITIH4_NPLVWVHASPEHVVVTR_vs_IBP3_YGQPLPGYTTK
113 и 100
0,030
0,682
CATD_VGFAEAAR_vs_IGF2_GIVEECCFR
57 и 103
0,012
0,711
ITIH4_NPLVWVHASPEHVVVTR_vs_PSG3_VSAPSGTGHLPGLNPL
113 и 134
0,033
0,678
CATD_VGFAEAAR_vs_ITIH4_ILDDLSPR
57 и 112
0,002
0,765
ITIH4_NPLVWVHASPEHVVVTR_vs_SOM2.CSH_NYGLLYCFR
113 и 138
0,021
0,693
CATD_VGFAEAAR_vs_LYAM1_SYYWIGIR
57 и 120
0,000
0,807
ITIH4_NPLVWVHASPEHVVVTR_vs_SPRL1_VLTHSELAPLR
113 и 140
0,042
0,670
CATD_VGFAEAAR_vs_NCAM1_GLGEISAASEFK
57 и 121
0,014
0,706
ITIH4_NPLVWVHASPEHVVVTR_vs_TENX_LSQLSVTDVTTSSLR
113 и 142
0,028
0,684
CATD_VGFAEAAR_vs_PGRP2_AGLLRPDYALLGHR
57 и 126
0,000
0,811
ITIH4_QLGLPGPPDVPDHAAYHPF_vs_TENX_LSQLSVTDVTTSSLR
114 и 142
0,029
0,683
CATD_VGFAEAAR_vs_PSG1_FQLPGQK
57 и 131
0,008
0,723
KNG1_DIPTNSPELEETLTHTITK_vs_SHBG_IALGGLLFPASNLR
116 и 18
0,040
0,672
CATD_VGFAEAAR_vs_PSG3_VSAPSGTGHLPGLNPL
57 и 134
0,014
0,706
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,009
0,717
CATD_VGFAEAAR_vs_SHBG_IALGGLLFPASNLR
57 и 18
0,001
0,787
KNG1_QVVAGLNFR_vs_PGRP2_AGLLRPDYALLGHR
117 и 126
0,020
0,695
CATD_VGFAEAAR_vs_SOM2.CSH_NYGLLYCFR
57 и 138
0,003
0,751
KNG1_QVVAGLNFR_vs_SHBG_IALGGLLFPASNLR
117 и 18
0,012
0,711
CATD_VGFAEAAR_vs_SOM2.CSH_SVEGSCGF
57 и 139
0,002
0,753
PAPP1_DIPHWLNPTR_vs_CRIS3_AVSPPAR
122 и 78
0,039
0,672
CATD_VGFAEAAR_vs_SPRL1_VLTHSELAPLR
57 и 140
0,003
0,746
PAPP1_DIPHWLNPTR_vs_CRIS3_YEDLYSNCK
122 и 79
0,035
0,676
CATD_VGFAEAAR_vs_TENX_LNWEAPPGAFDSFLLR
57 и 141
0,003
0,745
PAPP1_DIPHWLNPTR_vs_CSH_AHQLAIDTYQEFEETYIPK
122 и 80
0,029
0,683
CATD_VGFAEAAR_vs_TENX_LSQLSVTDVTTSSLR
57 и 142
0,013
0,708
PAPP1_DIPHWLNPTR_vs_CSH_ISLLLIESWLEPVR
122 и 81
0,019
0,696
CATD_VGFAEAAR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
57 и 144
0,005
0,735
PAPP1_DIPHWLNPTR_vs_IBP1_VVESLAK
122 и 97
0,047
0,666
CATD_VGFAEAAR_vs_VTDB_ELPEHTVK
57 и 147
0,004
0,743
PAPP1_DIPHWLNPTR_vs_IBP2_LIQGAPTIR
122 и 98
0,027
0,686
CATD_VSTLPAITLK_vs_ALS_IRPHTFTGLSGLR
58 и 40
0,002
0,761
PAPP1_DIPHWLNPTR_vs_ITIH4_ILDDLSPR
122 и 112
0,046
0,667
CATD_VSTLPAITLK_vs_C163A_INPASLDK
58 и 54
0,002
0,758
PAPP1_DIPHWLNPTR_vs_LYAM1_SYYWIGIR
122 и 120
0,007
0,726
CATD_VSTLPAITLK_vs_CRIS3_AVSPPAR
58 и 78
0,006
0,731
PAPP1_DIPHWLNPTR_vs_PGRP2_AGLLRPDYALLGHR
122 и 126
0,007
0,725
CATD_VSTLPAITLK_vs_CRIS3_YEDLYSNCK
58 и 79
0,004
0,741
PAPP1_DIPHWLNPTR_vs_PRG2_WNFAYWAAHQPWSR
122 и 129
0,021
0,693
CATD_VSTLPAITLK_vs_CSH_AHQLAIDTYQEFEETYIPK
58 и 80
0,001
0,775
PAPP1_DIPHWLNPTR_vs_PSG1_FQLPGQK
122 и 131
0,043
0,670
CATD_VSTLPAITLK_vs_CSH_ISLLLIESWLEPVR
58 и 81
0,001
0,769
PAPP1_DIPHWLNPTR_vs_SHBG_IALGGLLFPASNLR
122 и 18
0,005
0,734
CATD_VSTLPAITLK_vs_FBLN1_TGYYFDGISR
58 и 86
0,022
0,692
PAPP1_DIPHWLNPTR_vs_SOM2.CSH_NYGLLYCFR
122 и 138
0,028
0,684
CATD_VSTLPAITLK_vs_IBP1_VVESLAK
58 и 97
0,006
0,730
PAPP1_DIPHWLNPTR_vs_SOM2.CSH_SVEGSCGF
122 и 139
0,004
0,743
CATD_VSTLPAITLK_vs_IBP2_LIQGAPTIR
58 и 98
0,011
0,714
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,008
0,722
CATD_VSTLPAITLK_vs_IBP3_FLNVLSPR
58 и 99
0,005
0,733
PEDF_LQSLFDSPDFSK_vs_PGRP2_AGLLRPDYALLGHR
124 и 126
0,024
0,689
CATD_VSTLPAITLK_vs_IBP3_YGQPLPGYTTK
58 и 100
0,006
0,729
PEDF_LQSLFDSPDFSK_vs_SHBG_IALGGLLFPASNLR
124 и 18
0,008
0,720
CATD_VSTLPAITLK_vs_IGF2_GIVEECCFR
58 и 103
0,004
0,744
PEDF_LQSLFDSPDFSK_vs_VTDB_ELPEHTVK
124 и 147
0,023
0,690
CATD_VSTLPAITLK_vs_ITIH4_ILDDLSPR
58 и 112
0,001
0,784
PEDF_TVQAVLTVPK_vs_LYAM1_SYYWIGIR
125 и 120
0,008
0,722
CATD_VSTLPAITLK_vs_LYAM1_SYYWIGIR
58 и 120
0,000
0,825
PEDF_TVQAVLTVPK_vs_PGRP2_AGLLRPDYALLGHR
125 и 126
0,019
0,697
CATD_VSTLPAITLK_vs_NCAM1_GLGEISAASEFK
58 и 121
0,006
0,728
PEDF_TVQAVLTVPK_vs_SHBG_IALGGLLFPASNLR
125 и 18
0,007
0,726
CATD_VSTLPAITLK_vs_PGRP2_AGLLRPDYALLGHR
58 и 126
0,000
0,841
PRDX2_GLFIIDGK_vs_LYAM1_SYYWIGIR
128 и 120
0,034
0,677
CATD_VSTLPAITLK_vs_PSG1_FQLPGQK
58 и 131
0,016
0,701
PRDX2_GLFIIDGK_vs_PGRP2_AGLLRPDYALLGHR
128 и 126
0,026
0,687
CATD_VSTLPAITLK_vs_PSG3_VSAPSGTGHLPGLNPL
58 и 134
0,005
0,734
PRDX2_GLFIIDGK_vs_SHBG_IALGGLLFPASNLR
128 и 18
0,011
0,713
CATD_VSTLPAITLK_vs_SHBG_IALGGLLFPASNLR
58 и 18
0,000
0,810
PSG2_IHPSYTNYR_vs_IBP2_LIQGAPTIR
133 и 98
0,038
0,674
CATD_VSTLPAITLK_vs_SOM2.CSH_NYGLLYCFR
58 и 138
0,003
0,745
PSG2_IHPSYTNYR_vs_LYAM1_SYYWIGIR
133 и 120
0,023
0,690
CATD_VSTLPAITLK_vs_SOM2.CSH_SVEGSCGF
58 и 139
0,004
0,741
PSG2_IHPSYTNYR_vs_PGRP2_AGLLRPDYALLGHR
133 и 126
0,031
0,680
CATD_VSTLPAITLK_vs_SPRL1_VLTHSELAPLR
58 и 140
0,002
0,759
PSG2_IHPSYTNYR_vs_SHBG_IALGGLLFPASNLR
133 и 18
0,009
0,717
CATD_VSTLPAITLK_vs_TENX_LNWEAPPGAFDSFLLR
58 и 141
0,001
0,780
PTGDS_GPGEDFR_vs_LYAM1_SYYWIGIR
137 и 120
0,006
0,728
CATD_VSTLPAITLK_vs_TENX_LSQLSVTDVTTSSLR
58 и 142
0,005
0,736
PTGDS_GPGEDFR_vs_PGRP2_AGLLRPDYALLGHR
137 и 126
0,006
0,731
CATD_VSTLPAITLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
58 и 144
0,002
0,753
PTGDS_GPGEDFR_vs_SHBG_IALGGLLFPASNLR
137 и 18
0,009
0,719
CATD_VSTLPAITLK_vs_VTDB_ELPEHTVK
58 и 147
0,001
0,778
PTGDS_GPGEDFR_vs_SOM2.CSH_SVEGSCGF
137 и 139
0,027
0,685
CBPN_EALIQFLEQVHQGIK_vs_SHBG_IALGGLLFPASNLR
59 и 18
0,023
0,690
THBG_AVLHIGEK_vs_PGRP2_AGLLRPDYALLGHR
143 и 126
0,047
0,666
CBPN_NNANGVDLNR_vs_LYAM1_SYYWIGIR
60 и 120
0,038
0,674
THBG_AVLHIGEK_vs_SHBG_IALGGLLFPASNLR
143 и 18
0,039
0,673
CBPN_NNANGVDLNR_vs_SHBG_IALGGLLFPASNLR
60 и 18
0,016
0,702
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,009
0,717
CD14_LTVGAAQVPAQLLVGALR_vs_LYAM1_SYYWIGIR
61 и 120
0,008
0,721
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,012
0,711
CD14_LTVGAAQVPAQLLVGALR_vs_PGRP2_AGLLRPDYALLGHR
61 и 126
0,005
0,735
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,011
0,714
CD14_LTVGAAQVPAQLLVGALR_vs_SHBG_IALGGLLFPASNLR
61 и 18
0,006
0,731
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,023
0,690
CD14_LTVGAAQVPAQLLVGALR_vs_VTDB_ELPEHTVK
61 и 147
0,041
0,671
VTNC_VDTVDPPYPR_vs_PGRP2_AGLLRPDYALLGHR
150 и 126
0,024
0,688
CD14_SWLAELQQWLKPGLK_vs_LYAM1_SYYWIGIR
62 и 120
0,015
0,704
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,008
0,722
CD14_SWLAELQQWLKPGLK_vs_PGRP2_AGLLRPDYALLGHR
62 и 126
0,011
0,712
Таблица 59: дополнительные обращения
Обращение
SEQ ID №
Обращение
SEQ ID №
AFAM_DADPDTFFAK_vs_ALS_IRPHTFTGLSGLR
37 и 40
HABP2_FLNWIK_vs_IBP1_VVESLAK
92 и 97
AFAM_DADPDTFFAK_vs_CRIS3_AVSPPAR
37 и 78
HEMO_NFPSPVDAAFR_vs_IBP1_VVESLAK
93 и 97
AFAM_DADPDTFFAK_vs_CSH_AHQLAIDTYQEFEETYIPK
37 и 80
HEMO_NFPSPVDAAFR_vs_PSG9_LFIPQITR
93 и 136
AFAM_DADPDTFFAK_vs_CSH_ISLLLIESWLEPVR
37 и 81
HEMO_NFPSPVDAAFR_vs_SPRL1_VLTHSELAPLR
93 и 140
AFAM_DADPDTFFAK_vs_IBP1_VVESLAK
37 и 97
HLACI_WAAVVVPSGEEQR_vs_IBP1_VVESLAK
95 и 97
AFAM_HFQNLGK_vs_C163A_INPASLDK
38 и 54
HLACI_WAAVVVPSGEEQR_vs_LYAM1_SYYWIGIR
95 и 120
AFAM_HFQNLGK_vs_CSH_AHQLAIDTYQEFEETYIPK
38 и 80
IBP6_GAQTLYVPNCDHR_vs_ALS_IRPHTFTGLSGLR
101 и 40
AFAM_HFQNLGK_vs_CSH_ISLLLIESWLEPVR
38 и 81
IBP6_GAQTLYVPNCDHR_vs_CRIS3_AVSPPAR
101 и 78
AFAM_HFQNLGK_vs_FBLN1_TGYYFDGISR
38 и 86
IBP6_GAQTLYVPNCDHR_vs_IBP1_VVESLAK
101 и 97
ANGT_DPTFIPAPIQAK_vs_ALS_IRPHTFTGLSGLR
42 и 40
IBP6_GAQTLYVPNCDHR_vs_IBP2_LIQGAPTIR
101 и 98
ANGT_DPTFIPAPIQAK_vs_IBP1_VVESLAK
42 и 97
IBP6_GAQTLYVPNCDHR_vs_IBP3_FLNVLSPR
101 и 99
ANGT_DPTFIPAPIQAK_vs_ITIH4_ILDDLSPR
42 и 112
IBP6_GAQTLYVPNCDHR_vs_IBP3_YGQPLPGYTTK
101 и 100
APOH_ATVVYQGER_vs_CSH_ISLLLIESWLEPVR
48 и 81
IBP6_GAQTLYVPNCDHR_vs_SPRL1_VLTHSELAPLR
101 и 140
APOH_ATVVYQGER_vs_IBP2_LIQGAPTIR
48 и 98
IBP6_GAQTLYVPNCDHR_vs_TENX_LNWEAPPGAFDSFLLR
101 и 141
APOH_ATVVYQGER_vs_SOM2.CSH_SVEGSCGF
48 и 139
IBP6_GAQTLYVPNCDHR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
101 и 144
B2MG_VEHSDLSFSK_vs_C163A_INPASLDK
50 и 54
IBP6_HLDSVLQQLQTEVYR_vs_CRIS3_AVSPPAR
102 и 78
B2MG_VEHSDLSFSK_vs_IBP1_VVESLAK
50 и 97
IBP6_HLDSVLQQLQTEVYR_vs_IBP1_VVESLAK
102 и 97
B2MG_VEHSDLSFSK_vs_PSG9_LFIPQITR
50 и 136
IBP6_HLDSVLQQLQTEVYR_vs_IBP2_LIQGAPTIR
102 и 98
B2MG_VEHSDLSFSK_vs_SPRL1_VLTHSELAPLR
50 и 140
IBP6_HLDSVLQQLQTEVYR_vs_IBP3_FLNVLSPR
102 и 99
B2MG_VNHVTLSQPK_vs_CSH_AHQLAIDTYQEFEETYIPK
51 и 80
IBP6_HLDSVLQQLQTEVYR_vs_IBP3_YGQPLPGYTTK
102 и 100
B2MG_VNHVTLSQPK_vs_FBLN1_TGYYFDGISR
51 и 86
IBP6_HLDSVLQQLQTEVYR_vs_PSG9_LFIPQITR
102 и 136
B2MG_VNHVTLSQPK_vs_PSG9_LFIPQITR
51 и 136
IBP6_HLDSVLQQLQTEVYR_vs_SOM2.CSH_SVEGSCGF
102 и 139
B2MG_VNHVTLSQPK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
51 и 144
IBP6_HLDSVLQQLQTEVYR_vs_SPRL1_VLTHSELAPLR
102 и 140
B2MG_VNHVTLSQPK_vs_VTDB_ELPEHTVK
51 и 147
IBP6_HLDSVLQQLQTEVYR_vs_TENX_LNWEAPPGAFDSFLLR
102 и 141
BGH3_LTLLAPLNSVFK_vs_C163A_INPASLDK
52 и 54
IBP6_HLDSVLQQLQTEVYR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
102 и 144
BGH3_LTLLAPLNSVFK_vs_IBP1_VVESLAK
52 и 97
INHBC_LDFHFSSDR_vs_IBP1_VVESLAK
107 и 97
BGH3_LTLLAPLNSVFK_vs_SPRL1_VLTHSELAPLR
52 и 140
ITIH3_ALDLSLK_vs_PSG9_LFIPQITR
111 и 136
BGH3_LTLLAPLNSVFK_vs_VTDB_ELPEHTVK
52 и 147
ITIH4_NPLVWVHASPEHVVVTR_vs_C163A_INPASLDK
113 и 54
CAH1_GGPFSDSYR_vs_ALS_IRPHTFTGLSGLR
56 и 40
ITIH4_NPLVWVHASPEHVVVTR_vs_CSH_ISLLLIESWLEPVR
113 и 81
CAH1_GGPFSDSYR_vs_CSH_AHQLAIDTYQEFEETYIPK
56 и 80
ITIH4_NPLVWVHASPEHVVVTR_vs_PSG1_FQLPGQK
113 и 131
CAH1_GGPFSDSYR_vs_CSH_ISLLLIESWLEPVR
56 и 81
ITIH4_QLGLPGPPDVPDHAAYHPF_vs_ALS_IRPHTFTGLSGLR
114 и 40
CAH1_GGPFSDSYR_vs_FBLN1_TGYYFDGISR
56 и 86
ITIH4_QLGLPGPPDVPDHAAYHPF_vs_C163A_INPASLDK
114 и 54
CAH1_GGPFSDSYR_vs_IBP2_LIQGAPTIR
56 и 98
ITIH4_QLGLPGPPDVPDHAAYHPF_vs_CHL1_VIAVNEVGR
114 и 66
CAH1_GGPFSDSYR_vs_IBP3_YGQPLPGYTTK
56 и 100
ITIH4_QLGLPGPPDVPDHAAYHPF_vs_CSH_AHQLAIDTYQEFEETYIPK
114 и 80
CAH1_GGPFSDSYR_vs_IGF2_GIVEECCFR
56 и 103
ITIH4_QLGLPGPPDVPDHAAYHPF_vs_FBLN1_TGYYFDGISR
114 и 86
CAH1_GGPFSDSYR_vs_ITIH4_ILDDLSPR
56 и 112
ITIH4_QLGLPGPPDVPDHAAYHPF_vs_IBP3_FLNVLSPR
114 и 99
CAH1_GGPFSDSYR_vs_PRG2_WNFAYWAAHQPWSR
56 и 129
ITIH4_QLGLPGPPDVPDHAAYHPF_vs_IBP3_YGQPLPGYTTK
114 и 100
CAH1_GGPFSDSYR_vs_SOM2.CSH_SVEGSCGF
56 и 139
ITIH4_QLGLPGPPDVPDHAAYHPF_vs_IGF2_GIVEECCFR
114 и 103
CAH1_GGPFSDSYR_vs_SPRL1_VLTHSELAPLR
56 и 140
ITIH4_QLGLPGPPDVPDHAAYHPF_vs_ITIH4_ILDDLSPR
114 и 112
CAH1_GGPFSDSYR_vs_VTDB_ELPEHTVK
56 и 147
ITIH4_QLGLPGPPDVPDHAAYHPF_vs_NCAM1_GLGEISAASEFK
114 и 121
CBPN_EALIQFLEQVHQGIK_vs_CRIS3_AVSPPAR
59 и 78
ITIH4_QLGLPGPPDVPDHAAYHPF_vs_PRG2_WNFAYWAAHQPWSR
114 и 129
CBPN_EALIQFLEQVHQGIK_vs_IBP1_VVESLAK
59 и 97
ITIH4_QLGLPGPPDVPDHAAYHPF_vs_PSG1_FQLPGQK
114 и 131
CBPN_EALIQFLEQVHQGIK_vs_PGRP2_AGLLRPDYALLGHR
59 и 126
ITIH4_QLGLPGPPDVPDHAAYHPF_vs_SOM2.CSH_NYGLLYCFR
114 и 138
CBPN_EALIQFLEQVHQGIK_vs_SPRL1_VLTHSELAPLR
59 и 140
ITIH4_QLGLPGPPDVPDHAAYHPF_vs_SPRL1_VLTHSELAPLR
114 и 140
CBPN_NNANGVDLNR_vs_CRIS3_AVSPPAR
60 и 78
ITIH4_QLGLPGPPDVPDHAAYHPF_vs_TENX_LNWEAPPGAFDSFLLR
114 и 141
CBPN_NNANGVDLNR_vs_IBP1_VVESLAK
60 и 97
ITIH4_QLGLPGPPDVPDHAAYHPF_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
114 и 144
CBPN_NNANGVDLNR_vs_PSG9_LFIPQITR
60 и 136
ITIH4_QLGLPGPPDVPDHAAYHPF_vs_VTDB_ELPEHTVK
114 и 147
CD14_LTVGAAQVPAQLLVGALR_vs_ALS_IRPHTFTGLSGLR
61 и 40
KNG1_DIPTNSPELEETLTHTITK_vs_C163A_INPASLDK
116 и 54
CD14_LTVGAAQVPAQLLVGALR_vs_IBP2_LIQGAPTIR
61 и 98
KNG1_DIPTNSPELEETLTHTITK_vs_IBP3_FLNVLSPR
116 и 99
CD14_LTVGAAQVPAQLLVGALR_vs_SOM2.CSH_NYGLLYCFR
61 и 138
KNG1_DIPTNSPELEETLTHTITK_vs_IBP3_YGQPLPGYTTK
116 и 100
CD14_SWLAELQQWLKPGLK_vs_ALS_IRPHTFTGLSGLR
62 и 40
KNG1_DIPTNSPELEETLTHTITK_vs_PGRP2_AGLLRPDYALLGHR
116 и 126
CD14_SWLAELQQWLKPGLK_vs_CSH_ISLLLIESWLEPVR
62 и 81
KNG1_DIPTNSPELEETLTHTITK_vs_PSG9_LFIPQITR
116 и 136
CD14_SWLAELQQWLKPGLK_vs_IBP3_FLNVLSPR
62 и 99
KNG1_DIPTNSPELEETLTHTITK_vs_SOM2.CSH_SVEGSCGF
116 и 139
CD14_SWLAELQQWLKPGLK_vs_IBP3_YGQPLPGYTTK
62 и 100
KNG1_DIPTNSPELEETLTHTITK_vs_VTDB_ELPEHTVK
116 и 147
CD14_SWLAELQQWLKPGLK_vs_NCAM1_GLGEISAASEFK
62 и 121
KNG1_QVVAGLNFR_vs_ALS_IRPHTFTGLSGLR
117 и 40
CD14_SWLAELQQWLKPGLK_vs_PSG9_LFIPQITR
62 и 136
KNG1_QVVAGLNFR_vs_CSH_ISLLLIESWLEPVR
117 и 81
CD14_SWLAELQQWLKPGLK_vs_SOM2.CSH_NYGLLYCFR
62 и 138
KNG1_QVVAGLNFR_vs_PSG9_LFIPQITR
117 и 136
CFAB_YGLVTYATYPK_vs_IBP1_VVESLAK
64 и 97
KNG1_QVVAGLNFR_vs_SOM2.CSH_NYGLLYCFR
117 и 138
CLUS_ASSIIDELFQDR_vs_CSH_ISLLLIESWLEPVR
67 и 81
KNG1_QVVAGLNFR_vs_SOM2.CSH_SVEGSCGF
117 и 139
CLUS_ASSIIDELFQDR_vs_ITIH4_ILDDLSPR
67 и 112
LBP_ITGFLKPGK_vs_FBLN1_TGYYFDGISR
118 и 86
CLUS_ASSIIDELFQDR_vs_PRG2_WNFAYWAAHQPWSR
67 и 129
LBP_ITGFLKPGK_vs_NCAM1_GLGEISAASEFK
118 и 121
CLUS_ASSIIDELFQDR_vs_PSG9_LFIPQITR
67 и 136
PAPP1_DIPHWLNPTR_vs_ALS_IRPHTFTGLSGLR
122 и 40
CLUS_ASSIIDELFQDR_vs_SOM2.CSH_SVEGSCGF
67 и 139
PAPP1_DIPHWLNPTR_vs_CHL1_VIAVNEVGR
122 и 66
CLUS_ASSIIDELFQDR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
67 и 144
PAPP1_DIPHWLNPTR_vs_FBLN1_TGYYFDGISR
122 и 86
CLUS_LFDSDPITVTVPVEVSR_vs_CSH_ISLLLIESWLEPVR
68 и 81
PAPP1_DIPHWLNPTR_vs_IBP3_FLNVLSPR
122 и 99
CLUS_LFDSDPITVTVPVEVSR_vs_NCAM1_GLGEISAASEFK
68 и 121
PAPP1_DIPHWLNPTR_vs_IBP3_YGQPLPGYTTK
122 и 100
CLUS_LFDSDPITVTVPVEVSR_vs_PSG9_LFIPQITR
68 и 136
PAPP1_DIPHWLNPTR_vs_IGF2_GIVEECCFR
122 и 103
CLUS_LFDSDPITVTVPVEVSR_vs_SOM2.CSH_NYGLLYCFR
68 и 138
PAPP1_DIPHWLNPTR_vs_NCAM1_GLGEISAASEFK
122 и 121
CLUS_LFDSDPITVTVPVEVSR_vs_SOM2.CSH_SVEGSCGF
68 и 139
PAPP1_DIPHWLNPTR_vs_PSG3_VSAPSGTGHLPGLNPL
122 и 134
CO5_TLLPVSKPEIR_vs_C163A_INPASLDK
70 и 54
PAPP1_DIPHWLNPTR_vs_SPRL1_VLTHSELAPLR
122 и 140
CO5_TLLPVSKPEIR_vs_IBP1_VVESLAK
70 и 97
PAPP1_DIPHWLNPTR_vs_TENX_LNWEAPPGAFDSFLLR
122 и 141
CO5_TLLPVSKPEIR_vs_IBP3_FLNVLSPR
70 и 99
PAPP1_DIPHWLNPTR_vs_TENX_LSQLSVTDVTTSSLR
122 и 142
CO5_TLLPVSKPEIR_vs_PSG9_LFIPQITR
70 и 136
PAPP1_DIPHWLNPTR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
122 и 144
CO5_VFQFLEK_vs_IBP1_VVESLAK
71 и 97
PAPP1_DIPHWLNPTR_vs_VTDB_ELPEHTVK
122 и 147
CO5_VFQFLEK_vs_PSG9_LFIPQITR
71 и 136
PEDF_LQSLFDSPDFSK_vs_PSG9_LFIPQITR
124 и 136
CO5_VFQFLEK_vs_SPRL1_VLTHSELAPLR
71 и 140
PEDF_LQSLFDSPDFSK_vs_SPRL1_VLTHSELAPLR
124 и 140
CO6_ALNHLPLEYNSALYSR_vs_CRIS3_AVSPPAR
72 и 78
PEDF_TVQAVLTVPK_vs_IBP1_VVESLAK
125 и 97
CO6_ALNHLPLEYNSALYSR_vs_IBP1_VVESLAK
72 и 97
PEDF_TVQAVLTVPK_vs_IBP2_LIQGAPTIR
125 и 98
CO6_ALNHLPLEYNSALYSR_vs_PSG9_LFIPQITR
72 и 136
PEDF_TVQAVLTVPK_vs_IBP3_FLNVLSPR
125 и 99
CO6_ALNHLPLEYNSALYSR_vs_SPRL1_VLTHSELAPLR
72 и 140
PEDF_TVQAVLTVPK_vs_ITIH4_ILDDLSPR
125 и 112
CO8A_SLLQPNK_vs_CSH_ISLLLIESWLEPVR
74 и 81
PRDX2_GLFIIDGK_vs_ALS_IRPHTFTGLSGLR
128 и 40
CO8A_SLLQPNK_vs_ITIH4_ILDDLSPR
74 и 112
PRDX2_GLFIIDGK_vs_CSH_AHQLAIDTYQEFEETYIPK
128 и 80
CO8A_SLLQPNK_vs_SOM2.CSH_NYGLLYCFR
74 и 138
PRDX2_GLFIIDGK_vs_CSH_ISLLLIESWLEPVR
128 и 81
CO8A_SLLQPNK_vs_SOM2.CSH_SVEGSCGF
74 и 139
PRDX2_GLFIIDGK_vs_FBLN1_TGYYFDGISR
128 и 86
CO8B_QALEEFQK_vs_CSH_ISLLLIESWLEPVR
76 и 81
PRDX2_GLFIIDGK_vs_IBP3_FLNVLSPR
128 и 99
CO8B_QALEEFQK_vs_ITIH4_ILDDLSPR
76 и 112
PRDX2_GLFIIDGK_vs_IBP3_YGQPLPGYTTK
128 и 100
F13B_GDTYPAELYITGSILR_vs_ALS_IRPHTFTGLSGLR
84 и 40
PRDX2_GLFIIDGK_vs_IGF2_GIVEECCFR
128 и 103
F13B_GDTYPAELYITGSILR_vs_C163A_INPASLDK
84 и 54
PRDX2_GLFIIDGK_vs_ITIH4_ILDDLSPR
128 и 112
F13B_GDTYPAELYITGSILR_vs_CSH_ISLLLIESWLEPVR
84 и 81
PRDX2_GLFIIDGK_vs_NCAM1_GLGEISAASEFK
128 и 121
F13B_GDTYPAELYITGSILR_vs_ITIH4_ILDDLSPR
84 и 112
PRDX2_GLFIIDGK_vs_PSG1_FQLPGQK
128 и 131
F13B_GDTYPAELYITGSILR_vs_NCAM1_GLGEISAASEFK
84 и 121
PRDX2_GLFIIDGK_vs_SOM2.CSH_NYGLLYCFR
128 и 138
F13B_GDTYPAELYITGSILR_vs_PSG9_LFIPQITR
84 и 136
PRDX2_GLFIIDGK_vs_SOM2.CSH_SVEGSCGF
128 и 139
F13B_GDTYPAELYITGSILR_vs_SOM2.CSH_NYGLLYCFR
84 и 138
PSG11_LFIPQITPK_vs_LYAM1_SYYWIGIR
132 и 120
F13B_GDTYPAELYITGSILR_vs_SOM2.CSH_SVEGSCGF
84 и 139
PSG11_LFIPQITPK_vs_SHBG_IALGGLLFPASNLR
132 и 18
FBLN3_IPSNPSHR_vs_ALS_IRPHTFTGLSGLR
87 и 40
PSG2_IHPSYTNYR_vs_PRG2_WNFAYWAAHQPWSR
133 и 129
FBLN3_IPSNPSHR_vs_FBLN1_TGYYFDGISR
87 и 86
PTGDS_GPGEDFR_vs_CSH_AHQLAIDTYQEFEETYIPK
137 и 80
FBLN3_IPSNPSHR_vs_IBP3_FLNVLSPR
87 и 99
PTGDS_GPGEDFR_vs_CSH_ISLLLIESWLEPVR
137 и 81
FBLN3_IPSNPSHR_vs_ITIH4_ILDDLSPR
87 и 112
PTGDS_GPGEDFR_vs_IBP2_LIQGAPTIR
137 и 98
FBLN3_IPSNPSHR_vs_NCAM1_GLGEISAASEFK
87 и 121
PTGDS_GPGEDFR_vs_PSG9_LFIPQITR
137 и 136
FBLN3_IPSNPSHR_vs_PSG3_VSAPSGTGHLPGLNPL
87 и 134
THBG_AVLHIGEK_vs_CRIS3_AVSPPAR
143 и 78
FBLN3_IPSNPSHR_vs_PSG9_LFIPQITR
87 и 136
THBG_AVLHIGEK_vs_IBP1_VVESLAK
143 и 97
FBLN3_IPSNPSHR_vs_VTDB_ELPEHTVK
87 и 147
THBG_AVLHIGEK_vs_IBP3_FLNVLSPR
143 и 99
FETUA_FSVVYAK_vs_NCAM1_GLGEISAASEFK
88 и 121
THBG_AVLHIGEK_vs_PSG9_LFIPQITR
143 и 136
FETUA_HTLNQIDEVK_vs_NCAM1_GLGEISAASEFK
89 и 121
THBG_AVLHIGEK_vs_SPRL1_VLTHSELAPLR
143 и 140
HABP2_FLNWIK_vs_ALS_IRPHTFTGLSGLR
92 и 40
VTNC_VDTVDPPYPR_vs_IBP1_VVESLAK
150 и 97
Таблица 60: хронологические белки
Короткое название
Название белка
Анализируемое вещество
SEQ ID №
ADA12
Adam 12
FGFGGSTDSGPIR
151
ADA12
Adam 12
LIEIANHVDK
152
ANGT
Ангиотензиноген
DPTFIPAPIQAK
42
CSH
Гормон хорионический соматомаммотропин 1 и 2
AHQLAIDTYQEFEETYIPK
80
CSH
Гормон хорионический соматомаммотропин 1 и 2
ISLLLIESWLEPVR
81
FBLN1
Фибулин-1
TGYYFDGISR
86
PAI2
Ингибитор активатора плазминогена 2
LNIGYIEDLK
153
PAPP1
Паппализин-1
DIPHWLNPTR
122
PSG11
Специфичный β1-гликопротеин беременности 11
DLYHYITSYVVDGEIIIYGPAYSGR
130
PSG1
Специфичный β1-гликопротеин беременности 1
FQLPGQK
131
PSG2
Специфичный β1-гликопротеин беременности 2
IHPSYTNYR
133
PSG6
Специфичный β1-гликопротеин беременности 6
SNPVTLNVLYGPDLPR
154
PSG9
Специфичный β1-гликопротеин беременности 9
DVLLLVHNLPQNLPGYFWYK
135
PSG9
Специфичный β1-гликопротеин беременности 9
LFIPQITR
136
SOM2.CSH
Плацентарный гормон роста и гормон хорионический соматомаммотропин 1 и 2
NYGLLYCFR
138
SOM2.CSH
Плацентарный гормон роста и гормон хорионический соматомаммотропин 1 и 2
SVEGSCGF
139
TENX
Танасцин-X
LSQLSVTDVTTSSLR
142
TIE1
Рецептор тирозиновой протеинкиназы
VSWSLPLVPGPLVGDGFLLR
144
VGFR3
Рецептор фактора роста эндотелия сосудов 3
SGVDLADSNQK
155
VGFR3
Рецептор фактора роста эндотелия сосудов 3
HATLSLSIPR
156
Таблица 61: эффективность классификации по обращениям, недели 17-21.
AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, без BMI-стратификации, отдельно для PPROM и PTL.
Обращение
SEQ ID №
119_153_aBMI_37 PTL ROC_AUC
119_153_aBMI_37 PTL P-значение
119_153_aBMI_37 PPROM ROC_AUC
119_153_aBMI_37 PPROM P-значение
A2GL_DLLLPQPDLR_vs_SHBG_IALGGLLFPASNLR
34 и 18
0,56
0,18
0,66
0,001
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,58
0,0951
0,65
0,0019
AFAM_HFQNLGK_vs_IBP3_YGQPLPGYTTK
38 и 100
0,56
0,2369
0,66
0,0012
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,57
0,1185
0,67
5,00E-04
APOC3_GWVTDGFSSLK_vs_ALS_IRPHTFTGLSGLR
47 и 40
0,58
0,0739
0,66
8,00E-04
APOC3_GWVTDGFSSLK_vs_C163A_INPASLDK
47 и 54
0,58
0,0834
0,67
7,00E-04
APOC3_GWVTDGFSSLK_vs_CHL1_VIAVNEVGR
47 и 66
0,58
0,0919
0,66
0,0011
APOC3_GWVTDGFSSLK_vs_CRIS3_AVSPPAR
47 и 78
0,56
0,1948
0,68
2,00E-04
APOC3_GWVTDGFSSLK_vs_CRIS3_YEDLYSNCK
47 и 79
0,57
0,1492
0,68
2,00E-04
APOC3_GWVTDGFSSLK_vs_CSH_AHQLAIDTYQEFEETYIPK
47 и 80
0,59
0,0705
0,66
7,00E-04
APOC3_GWVTDGFSSLK_vs_CSH_ISLLLIESWLEPVR
47 и 81
0,58
0,1112
0,65
0,0026
APOC3_GWVTDGFSSLK_vs_FBLN1_TGYYFDGISR
47 и 86
0,56
0,1845
0,68
3,00E-04
APOC3_GWVTDGFSSLK_vs_IBP2_LIQGAPTIR
47 и 98
0,54
0,3592
0,66
8,00E-04
APOC3_GWVTDGFSSLK_vs_IBP3_FLNVLSPR
47 и 99
0,6
0,0431
0,67
3,00E-04
APOC3_GWVTDGFSSLK_vs_IBP3_YGQPLPGYTTK
47 и 100
0,59
0,0487
0,67
4,00E-04
APOC3_GWVTDGFSSLK_vs_IGF2_GIVEECCFR
47 и 103
0,6
0,0289
0,68
2,00E-04
APOC3_GWVTDGFSSLK_vs_ITIH4_ILDDLSPR
47 и 112
0,59
0,0664
0,66
8,00E-04
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,57
0,1171
0,69
1,00E-04
APOC3_GWVTDGFSSLK_vs_NCAM1_GLGEISAASEFK
47 и 121
0,58
0,081
0,65
0,0014
APOC3_GWVTDGFSSLK_vs_PGRP2_AGLLRPDYALLGHR
47 и 126
0,58
0,0931
0,68
2,00E-04
APOC3_GWVTDGFSSLK_vs_PSG3_VSAPSGTGHLPGLNPL
47 и 134
0,63
0,0045
0,65
0,0014
APOC3_GWVTDGFSSLK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
47 и 135
0,56
0,2234
0,66
0,001
APOC3_GWVTDGFSSLK_vs_PSG9_LFIPQITR
47 и 136
0,55
0,2511
0,65
0,0018
APOC3_GWVTDGFSSLK_vs_SHBG_IALGGLLFPASNLR
47 и 18
0,59
0,0618
0,67
4,00E-04
APOC3_GWVTDGFSSLK_vs_SPRL1_VLTHSELAPLR
47 и 140
0,56
0,2251
0,66
7,00E-04
APOC3_GWVTDGFSSLK_vs_TENX_LNWEAPPGAFDSFLLR
47 и 141
0,58
0,0834
0,68
2,00E-04
APOC3_GWVTDGFSSLK_vs_TENX_LSQLSVTDVTTSSLR
47 и 142
0,58
0,0829
0,67
5,00E-04
APOC3_GWVTDGFSSLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
47 и 144
0,59
0,0703
0,67
4,00E-04
APOC3_GWVTDGFSSLK_vs_VTDB_ELPEHTVK
47 и 147
0,59
0,0703
0,67
4,00E-04
APOH_ATVVYQGER_vs_PSG3_VSAPSGTGHLPGLNPL
48 и 134
0,66
0,001
0,57
0,1723
B2MG_VNHVTLSQPK_vs_SHBG_IALGGLLFPASNLR
51 и 18
0,55
0,2492
0,67
6,00E-04
C1QB_VPGLYYFTYHASSR_vs_PGRP2_AGLLRPDYALLGHR
55 и 126
0,55
0,2632
0,65
0,0018
C1QB_VPGLYYFTYHASSR_vs_SHBG_IALGGLLFPASNLR
55 и 18
0,57
0,1178
0,66
9,00E-04
CATD_VGFAEAAR_vs_TENX_LNWEAPPGAFDSFLLR
57 и 141
0,55
0,3208
0,67
6,00E-04
CD14_LTVGAAQVPAQLLVGALR_vs_SHBG_IALGGLLFPASNLR
61 и 18
0,55
0,3012
0,66
7,00E-04
CD14_LTVGAAQVPAQLLVGALR_vs_TENX_LNWEAPPGAFDSFLLR
61 и 141
0,54
0,403
0,67
5,00E-04
CD14_SWLAELQQWLKPGLK_vs_SHBG_IALGGLLFPASNLR
62 и 18
0,55
0,2536
0,67
6,00E-04
CD14_SWLAELQQWLKPGLK_vs_TENX_LNWEAPPGAFDSFLLR
62 и 141
0,54
0,3953
0,67
6,00E-04
CFAB_YGLVTYATYPK_vs_PGRP2_AGLLRPDYALLGHR
64 и 126
0,57
0,1412
0,65
0,0023
CFAB_YGLVTYATYPK_vs_PSG3_VSAPSGTGHLPGLNPL
64 и 134
0,67
3,00E-04
0,59
0,0542
CFAB_YGLVTYATYPK_vs_SHBG_IALGGLLFPASNLR
64 и 18
0,6
0,0426
0,65
0,0024
CO5_TLLPVSKPEIR_vs_PSG3_VSAPSGTGHLPGLNPL
70 и 134
0,66
0,001
0,58
0,1098
CO5_TLLPVSKPEIR_vs_SHBG_IALGGLLFPASNLR
70 и 18
0,57
0,1453
0,65
0,0016
CO5_TLLPVSKPEIR_vs_TENX_LNWEAPPGAFDSFLLR
70 и 141
0,55
0,2704
0,65
0,0015
CO5_VFQFLEK_vs_PSG3_VSAPSGTGHLPGLNPL
71 и 134
0,65
0,0013
0,57
0,129
CO5_VFQFLEK_vs_SHBG_IALGGLLFPASNLR
71 и 18
0,57
0,1316
0,66
0,0013
CO5_VFQFLEK_vs_TENX_LNWEAPPGAFDSFLLR
71 и 141
0,56
0,2455
0,65
0,0018
CO8A_SLLQPNK_vs_PSG3_VSAPSGTGHLPGLNPL
74 и 134
0,65
0,002
0,58
0,1094
CO8A_SLLQPNK_vs_SHBG_IALGGLLFPASNLR
74 и 18
0,57
0,1375
0,65
0,0024
CO8A_SLLQPNK_vs_TENX_LNWEAPPGAFDSFLLR
74 и 141
0,58
0,1016
0,65
0,0021
CO8B_QALEEFQK_vs_PSG3_VSAPSGTGHLPGLNPL
76 и 134
0,65
0,002
0,59
0,0605
CO8B_QALEEFQK_vs_SHBG_IALGGLLFPASNLR
76 и 18
0,56
0,181
0,65
0,0017
CO8B_QALEEFQK_vs_TENX_LNWEAPPGAFDSFLLR
76 и 141
0,58
0,1026
0,66
0,0012
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_SHBG_IALGGLLFPASNLR
82 и 18
0,57
0,1566
0,67
5,00E-04
ENPP2_TYLHTYESEI_vs_LYAM1_SYYWIGIR
83 и 120
0,55
0,3336
0,66
8,00E-04
ENPP2_TYLHTYESEI_vs_SHBG_IALGGLLFPASNLR
83 и 18
0,55
0,2717
0,67
3,00E-04
FETUA_FSVVYAK_vs_IGF2_GIVEECCFR
88 и 103
0,59
0,0659
0,67
4,00E-04
FETUA_FSVVYAK_vs_SHBG_IALGGLLFPASNLR
88 и 18
0,54
0,4516
0,67
3,00E-04
FETUA_FSVVYAK_vs_TENX_LNWEAPPGAFDSFLLR
88 и 141
0,54
0,3979
0,68
2,00E-04
FETUA_HTLNQIDEVK_vs_SHBG_IALGGLLFPASNLR
89 и 18
0,54
0,3878
0,67
5,00E-04
FETUA_HTLNQIDEVK_vs_TENX_LNWEAPPGAFDSFLLR
89 и 141
0,55
0,3019
0,68
2,00E-04
HABP2_FLNWIK_vs_CHL1_VIAVNEVGR
92 и 66
0,56
0,2049
0,66
0,0013
HABP2_FLNWIK_vs_IGF2_GIVEECCFR
92 и 103
0,61
0,0245
0,65
0,0019
HABP2_FLNWIK_vs_SHBG_IALGGLLFPASNLR
92 и 18
0,55
0,26
0,66
8,00E-04
HABP2_FLNWIK_vs_TENX_LNWEAPPGAFDSFLLR
92 и 141
0,56
0,2393
0,68
2,00E-04
HABP2_FLNWIK_vs_TENX_LSQLSVTDVTTSSLR
92 и 142
0,54
0,3466
0,66
9,00E-04
HEMO_NFPSPVDAAFR_vs_SHBG_IALGGLLFPASNLR
93 и 18
0,56
0,2286
0,67
3,00E-04
HEMO_NFPSPVDAAFR_vs_TENX_LNWEAPPGAFDSFLLR
93 и 141
0,54
0,4212
0,67
5,00E-04
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,55
0,2684
0,65
0,0016
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,55
0,2523
0,66
9,00E-04
IBP4_QCHPALDGQR_vs_PGRP2_AGLLRPDYALLGHR
2 и 126
0,57
0,1643
0,69
1,00E-04
IBP4_QCHPALDGQR_vs_PSG3_VSAPSGTGHLPGLNPL
2 и 134
0,67
3,00E-04
0,61
0,0185
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,6
0,0371
0,67
3,00E-04
IBP4_QCHPALDGQR_vs_TENX_LNWEAPPGAFDSFLLR
2 и 141
0,57
0,1255
0,67
6,00E-04
INHBC_LDFHFSSDR_vs_CHL1_VIAVNEVGR
107 и 66
0,56
0,188
0,67
6,00E-04
INHBC_LDFHFSSDR_vs_IBP3_FLNVLSPR
107 и 99
0,56
0,243
0,68
3,00E-04
INHBC_LDFHFSSDR_vs_IBP3_YGQPLPGYTTK
107 и 100
0,56
0,188
0,67
3,00E-04
INHBC_LDFHFSSDR_vs_IGF2_GIVEECCFR
107 и 103
0,59
0,0635
0,69
1,00E-04
INHBC_LDFHFSSDR_vs_ITIH4_ILDDLSPR
107 и 112
0,54
0,3936
0,66
7,00E-04
INHBC_LDFHFSSDR_vs_LYAM1_SYYWIGIR
107 и 120
0,53
0,549
0,68
2,00E-04
INHBC_LDFHFSSDR_vs_PGRP2_AGLLRPDYALLGHR
107 и 126
0,54
0,3911
0,71
0
INHBC_LDFHFSSDR_vs_PSG3_VSAPSGTGHLPGLNPL
107 и 134
0,6
0,0281
0,67
4,00E-04
INHBC_LDFHFSSDR_vs_SHBG_IALGGLLFPASNLR
107 и 18
0,56
0,1922
0,68
3,00E-04
INHBC_LDFHFSSDR_vs_TENX_LNWEAPPGAFDSFLLR
107 и 141
0,56
0,2054
0,7
0
INHBC_LDFHFSSDR_vs_TENX_LSQLSVTDVTTSSLR
107 и 142
0,55
0,2724
0,67
4,00E-04
INHBC_LDFHFSSDR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
107 и 144
0,55
0,29
0,66
7,00E-04
INHBC_LDFHFSSDR_vs_VTDB_ELPEHTVK
107 и 147
0,55
0,3313
0,68
1,00E-04
ITIH3_ALDLSLK_vs_SHBG_IALGGLLFPASNLR
111 и 18
0,56
0,1969
0,69
1,00E-04
KNG1_DIPTNSPELEETLTHTITK_vs_SHBG_IALGGLLFPASNLR
116 и 18
0,55
0,3283
0,66
0,001
KNG1_QVVAGLNFR_vs_SHBG_IALGGLLFPASNLR
117 и 18
0,53
0,4899
0,67
3,00E-04
LBP_ITGFLKPGK_vs_CHL1_VIAVNEVGR
118 и 66
0,56
0,2357
0,65
0,0019
LBP_ITGFLKPGK_vs_CRIS3_YEDLYSNCK
118 и 79
0,54
0,3459
0,67
4,00E-04
LBP_ITGFLKPGK_vs_IGF2_GIVEECCFR
118 и 103
0,58
0,1007
0,65
0,0024
LBP_ITGFLKPGK_vs_LYAM1_SYYWIGIR
118 и 120
0,53
0,487
0,68
1,00E-04
LBP_ITGFLKPGK_vs_PGRP2_AGLLRPDYALLGHR
118 и 126
0,53
0,4842
0,68
2,00E-04
LBP_ITGFLKPGK_vs_PSG3_VSAPSGTGHLPGLNPL
118 и 134
0,63
0,0062
0,65
0,0024
LBP_ITGFLKPGK_vs_SHBG_IALGGLLFPASNLR
118 и 18
0,56
0,2049
0,69
1,00E-04
LBP_ITGFLKPGK_vs_TENX_LNWEAPPGAFDSFLLR
118 и 141
0,54
0,382
0,66
8,00E-04
LBP_ITGFLKPGK_vs_VTDB_ELPEHTVK
118 и 147
0,54
0,3672
0,66
8,00E-04
LBP_ITLPDFTGDLR_vs_C163A_INPASLDK
119 и 54
0,54
0,3746
0,68
3,00E-04
LBP_ITLPDFTGDLR_vs_CHL1_VIAVNEVGR
119 и 66
0,57
0,1308
0,67
5,00E-04
LBP_ITLPDFTGDLR_vs_CRIS3_AVSPPAR
119 и 78
0,55
0,3298
0,69
1,00E-04
LBP_ITLPDFTGDLR_vs_CRIS3_YEDLYSNCK
119 и 79
0,56
0,2228
0,69
1,00E-04
LBP_ITLPDFTGDLR_vs_CSH_AHQLAIDTYQEFEETYIPK
119 и 80
0,56
0,1911
0,65
0,0025
LBP_ITLPDFTGDLR_vs_IGF2_GIVEECCFR
119 и 103
0,6
0,0373
0,66
9,00E-04
LBP_ITLPDFTGDLR_vs_ITIH4_ILDDLSPR
119 и 112
0,54
0,3482
0,67
3,00E-04
LBP_ITLPDFTGDLR_vs_LYAM1_SYYWIGIR
119 и 120
0,55
0,3048
0,71
0
LBP_ITLPDFTGDLR_vs_NCAM1_GLGEISAASEFK
119 и 121
0,57
0,1553
0,65
0,0025
LBP_ITLPDFTGDLR_vs_PGRP2_AGLLRPDYALLGHR
119 и 126
0,55
0,3105
0,7
0
LBP_ITLPDFTGDLR_vs_PSG3_VSAPSGTGHLPGLNPL
119 и 134
0,64
0,0028
0,66
9,00E-04
LBP_ITLPDFTGDLR_vs_SHBG_IALGGLLFPASNLR
119 и 18
0,57
0,115
0,7
0
LBP_ITLPDFTGDLR_vs_SOM2.CSH_SVEGSCGF
119 и 139
0,57
0,1245
0,65
0,0027
LBP_ITLPDFTGDLR_vs_TENX_LNWEAPPGAFDSFLLR
119 и 141
0,56
0,2387
0,68
1,00E-04
LBP_ITLPDFTGDLR_vs_TENX_LSQLSVTDVTTSSLR
119 и 142
0,55
0,2921
0,67
6,00E-04
LBP_ITLPDFTGDLR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
119 и 144
0,56
0,1974
0,66
8,00E-04
LBP_ITLPDFTGDLR_vs_VTDB_ELPEHTVK
119 и 147
0,56
0,1703
0,69
1,00E-04
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,53
0,5622
0,68
2,00E-04
PEDF_LQSLFDSPDFSK_vs_SHBG_IALGGLLFPASNLR
124 и 18
0,55
0,2724
0,66
0,0012
PEDF_LQSLFDSPDFSK_vs_TENX_LNWEAPPGAFDSFLLR
124 и 141
0,55
0,2473
0,68
3,00E-04
PEDF_LQSLFDSPDFSK_vs_TENX_LSQLSVTDVTTSSLR
124 и 142
0,56
0,2393
0,65
0,0019
PEDF_TVQAVLTVPK_vs_IGF2_GIVEECCFR
125 и 103
0,56
0,2022
0,66
0,0012
PEDF_TVQAVLTVPK_vs_TENX_LNWEAPPGAFDSFLLR
125 и 141
0,54
0,403
0,68
1,00E-04
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,57
0,1428
0,67
5,00E-04
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,57
0,1518
0,68
3,00E-04
VTNC_GQYCYELDEK_vs_PSG3_VSAPSGTGHLPGLNPL
149 и 134
0,68
2,00E-04
0,61
0,0197
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,6
0,0293
0,68
2,00E-04
VTNC_GQYCYELDEK_vs_TENX_LNWEAPPGAFDSFLLR
149 и 141
0,57
0,1513
0,67
3,00E-04
VTNC_GQYCYELDEK_vs_TENX_LSQLSVTDVTTSSLR
149 и 142
0,56
0,1901
0,65
0,0023
VTNC_GQYCYELDEK_vs_VTDB_ELPEHTVK
149 и 147
0,6
0,029
0,66
0,0011
VTNC_VDTVDPPYPR_vs_CHL1_VIAVNEVGR
150 и 66
0,59
0,0722
0,65
0,0025
VTNC_VDTVDPPYPR_vs_IGF2_GIVEECCFR
150 и 103
0,62
0,0142
0,65
0,0023
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,57
0,1416
0,68
2,00E-04
VTNC_VDTVDPPYPR_vs_PGRP2_AGLLRPDYALLGHR
150 и 126
0,55
0,2479
0,68
2,00E-04
VTNC_VDTVDPPYPR_vs_PSG3_VSAPSGTGHLPGLNPL
150 и 134
0,67
5,00E-04
0,62
0,0164
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,59
0,0644
0,68
2,00E-04
VTNC_VDTVDPPYPR_vs_TENX_LNWEAPPGAFDSFLLR
150 и 141
0,56
0,1805
0,67
4,00E-04
VTNC_VDTVDPPYPR_vs_VTDB_ELPEHTVK
150 и 147
0,61
0,0248
0,68
2,00E-04
Таблица 62: эффективность классификации по обращениям, недели 17-21.
AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, с BMI-стратификацией (>22≤37), отдельно для PPROM и PTL.
Обращение
SEQ ID №
119_153_rBMI_37 PTL ROC_AUC
119_153_rBMI_37 PTL P-значение
119_153_rBMI_37 PPROM ROC_AUC
119_153_rBMI_37 PPROM P-значение
A2GL_DLLLPQPDLR_vs_IGF2_GIVEECCFR
34 и 103
0,65
0,0065
0,58
0,195
A2GL_DLLLPQPDLR_vs_LYAM1_SYYWIGIR
34 и 120
0,61
0,0376
0,65
0,015
A2GL_DLLLPQPDLR_vs_PGRP2_AGLLRPDYALLGHR
34 и 126
0,57
0,1794
0,65
0,0119
A2GL_DLLLPQPDLR_vs_SHBG_IALGGLLFPASNLR
34 и 18
0,58
0,153
0,65
0,0106
AFAM_DADPDTFFAK_vs_IBP3_FLNVLSPR
37 и 99
0,57
0,2071
0,65
0,0131
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,61
0,0383
0,66
0,0068
AFAM_HFQNLGK_vs_IBP3_FLNVLSPR
38 и 99
0,56
0,2409
0,69
0,0019
AFAM_HFQNLGK_vs_IBP3_YGQPLPGYTTK
38 и 100
0,61
0,0525
0,67
0,0047
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,61
0,0559
0,68
0,0034
ANGT_DPTFIPAPIQAK_vs_CRIS3_AVSPPAR
42 и 78
0,57
0,1892
0,67
0,0053
ANGT_DPTFIPAPIQAK_vs_CRIS3_YEDLYSNCK
42 и 79
0,58
0,1223
0,66
0,0082
ANGT_DPTFIPAPIQAK_vs_LYAM1_SYYWIGIR
42 и 120
0,54
0,4394
0,7
0,0011
ANGT_DPTFIPAPIQAK_vs_PGRP2_AGLLRPDYALLGHR
42 и 126
0,53
0,6034
0,7
0,0011
ANGT_DPTFIPAPIQAK_vs_SHBG_IALGGLLFPASNLR
42 и 18
0,55
0,371
0,72
3,00E-04
ANGT_DPTFIPAPIQAK_vs_TENX_LNWEAPPGAFDSFLLR
42 и 141
0,55
0,4081
0,68
0,0023
APOC3_GWVTDGFSSLK_vs_C163A_INPASLDK
47 и 54
0,59
0,1051
0,68
0,0034
APOC3_GWVTDGFSSLK_vs_CRIS3_AVSPPAR
47 и 78
0,6
0,0815
0,67
0,006
APOC3_GWVTDGFSSLK_vs_CRIS3_YEDLYSNCK
47 и 79
0,61
0,0525
0,66
0,0066
APOC3_GWVTDGFSSLK_vs_IBP3_FLNVLSPR
47 и 99
0,61
0,0538
0,65
0,0142
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,6
0,082
0,67
0,004
APOC3_GWVTDGFSSLK_vs_PGRP2_AGLLRPDYALLGHR
47 и 126
0,58
0,1338
0,66
0,0079
APOC3_GWVTDGFSSLK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
47 и 135
0,57
0,223
0,67
0,0046
APOC3_GWVTDGFSSLK_vs_PSG9_LFIPQITR
47 и 136
0,55
0,3213
0,66
0,0088
APOC3_GWVTDGFSSLK_vs_SHBG_IALGGLLFPASNLR
47 и 18
0,59
0,1185
0,65
0,0145
APOH_ATVVYQGER_vs_IGF2_GIVEECCFR
48 и 103
0,65
0,0057
0,57
0,2705
BGH3_LTLLAPLNSVFK_vs_LYAM1_SYYWIGIR
52 и 120
0,57
0,1883
0,67
0,0059
BGH3_LTLLAPLNSVFK_vs_SHBG_IALGGLLFPASNLR
52 и 18
0,54
0,4803
0,68
0,0032
BGH3_LTLLAPLNSVFK_vs_TENX_LNWEAPPGAFDSFLLR
52 и 141
0,56
0,2834
0,66
0,0062
C1QB_VPGLYYFTYHASSR_vs_PGRP2_AGLLRPDYALLGHR
55 и 126
0,58
0,1331
0,66
0,0068
C1QB_VPGLYYFTYHASSR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
55 и 135
0,54
0,4346
0,68
0,0027
C1QB_VPGLYYFTYHASSR_vs_SHBG_IALGGLLFPASNLR
55 и 18
0,59
0,1159
0,65
0,0102
CFAB_YGLVTYATYPK_vs_CHL1_VIAVNEVGR
64 и 66
0,65
0,0075
0,56
0,3512
CFAB_YGLVTYATYPK_vs_IBP3_YGQPLPGYTTK
64 и 100
0,66
0,0032
0,55
0,4332
CFAB_YGLVTYATYPK_vs_IGF2_GIVEECCFR
64 и 103
0,67
0,0019
0,57
0,2156
CFAB_YGLVTYATYPK_vs_LYAM1_SYYWIGIR
64 и 120
0,65
0,008
0,6
0,1111
CFAB_YGLVTYATYPK_vs_PSG3_VSAPSGTGHLPGLNPL
64 и 134
0,7
3,00E-04
0,52
0,7399
CFAB_YGLVTYATYPK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
64 и 144
0,67
0,0026
0,56
0,2866
CO5_TLLPVSKPEIR_vs_IGF2_GIVEECCFR
70 и 103
0,66
0,0034
0,56
0,345
CO5_TLLPVSKPEIR_vs_PSG3_VSAPSGTGHLPGLNPL
70 и 134
0,69
4,00E-04
0,52
0,7583
CO5_TLLPVSKPEIR_vs_SHBG_IALGGLLFPASNLR
70 и 18
0,59
0,1116
0,65
0,0156
CO5_VFQFLEK_vs_IGF2_GIVEECCFR
71 и 103
0,67
0,0024
0,53
0,6116
CO5_VFQFLEK_vs_PSG3_VSAPSGTGHLPGLNPL
71 и 134
0,69
6,00E-04
0,5
0,994
CO6_ALNHLPLEYNSALYSR_vs_IGF2_GIVEECCFR
72 и 103
0,65
0,0073
0,57
0,2475
CO6_ALNHLPLEYNSALYSR_vs_PGRP2_AGLLRPDYALLGHR
72 и 126
0,55
0,3239
0,66
0,0075
CO8A_SLLQPNK_vs_IGF2_GIVEECCFR
74 и 103
0,67
0,0017
0,54
0,5019
CO8B_QALEEFQK_vs_IGF2_GIVEECCFR
76 и 103
0,66
0,0044
0,57
0,2487
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_LYAM1_SYYWIGIR
82 и 120
0,58
0,1424
0,67
0,0051
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_SHBG_IALGGLLFPASNLR
82 и 18
0,58
0,1601
0,68
0,0033
ENPP2_TYLHTYESEI_vs_LYAM1_SYYWIGIR
83 и 120
0,57
0,2271
0,66
0,0091
ENPP2_TYLHTYESEI_vs_SHBG_IALGGLLFPASNLR
83 и 18
0,55
0,3581
0,67
0,0044
HABP2_FLNWIK_vs_IGF2_GIVEECCFR
92 и 103
0,65
0,0059
0,62
0,0489
HABP2_FLNWIK_vs_LYAM1_SYYWIGIR
92 и 120
0,56
0,3019
0,67
0,0045
HABP2_FLNWIK_vs_TENX_LNWEAPPGAFDSFLLR
92 и 141
0,56
0,2726
0,67
0,0059
IBP4_QCHPALDGQR_vs_CRIS3_AVSPPAR
2 и 78
0,59
0,1197
0,66
0,0063
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,61
0,0378
0,66
0,0073
IBP4_QCHPALDGQR_vs_IBP3_YGQPLPGYTTK
2 и 100
0,66
0,0034
0,58
0,1711
IBP4_QCHPALDGQR_vs_IGF2_GIVEECCFR
2 и 103
0,67
0,0015
0,61
0,0651
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,61
0,0451
0,68
0,0032
IBP4_QCHPALDGQR_vs_PGRP2_AGLLRPDYALLGHR
2 и 126
0,58
0,1417
0,71
6,00E-04
IBP4_QCHPALDGQR_vs_PSG3_VSAPSGTGHLPGLNPL
2 и 134
0,68
0,0014
0,56
0,3528
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,6
0,0834
0,68
0,0024
INHBC_LDFHFSSDR_vs_ALS_IRPHTFTGLSGLR
107 и 40
0,56
0,2846
0,68
0,0026
INHBC_LDFHFSSDR_vs_CHL1_VIAVNEVGR
107 и 66
0,58
0,1366
0,66
0,0088
INHBC_LDFHFSSDR_vs_CRIS3_AVSPPAR
107 и 78
0,55
0,3173
0,67
0,006
INHBC_LDFHFSSDR_vs_CRIS3_YEDLYSNCK
107 и 79
0,57
0,2179
0,67
0,0043
INHBC_LDFHFSSDR_vs_IBP3_FLNVLSPR
107 и 99
0,59
0,1086
0,69
0,0015
INHBC_LDFHFSSDR_vs_IBP3_YGQPLPGYTTK
107 и 100
0,61
0,0439
0,67
0,004
INHBC_LDFHFSSDR_vs_IGF2_GIVEECCFR
107 и 103
0,63
0,0188
0,67
0,0039
INHBC_LDFHFSSDR_vs_ITIH4_ILDDLSPR
107 и 112
0,54
0,441
0,68
0,0031
INHBC_LDFHFSSDR_vs_LYAM1_SYYWIGIR
107 и 120
0,55
0,3239
0,69
0,0021
INHBC_LDFHFSSDR_vs_PGRP2_AGLLRPDYALLGHR
107 и 126
0,56
0,3109
0,7
9,00E-04
INHBC_LDFHFSSDR_vs_SHBG_IALGGLLFPASNLR
107 и 18
0,57
0,2071
0,68
0,0029
INHBC_LDFHFSSDR_vs_SPRL1_VLTHSELAPLR
107 и 140
0,57
0,21
0,68
0,0034
INHBC_LDFHFSSDR_vs_TENX_LNWEAPPGAFDSFLLR
107 и 141
0,59
0,1074
0,7
0,001
INHBC_LDFHFSSDR_vs_TENX_LSQLSVTDVTTSSLR
107 и 142
0,58
0,1633
0,66
0,0078
INHBC_LDFHFSSDR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
107 и 144
0,56
0,2388
0,67
0,0056
INHBC_LDFHFSSDR_vs_VTDB_ELPEHTVK
107 и 147
0,56
0,2531
0,71
6,00E-04
ITIH3_ALDLSLK_vs_SHBG_IALGGLLFPASNLR
111 и 18
0,58
0,1338
0,67
0,0042
KNG1_DIPTNSPELEETLTHTITK_vs_LYAM1_SYYWIGIR
116 и 120
0,56
0,3147
0,67
0,005
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,56
0,2882
0,67
0,0055
LBP_ITGFLKPGK_vs_CRIS3_AVSPPAR
118 и 78
0,56
0,2822
0,65
0,012
LBP_ITGFLKPGK_vs_CRIS3_YEDLYSNCK
118 и 79
0,57
0,1725
0,65
0,0135
LBP_ITGFLKPGK_vs_LYAM1_SYYWIGIR
118 и 120
0,56
0,307
0,67
0,004
LBP_ITGFLKPGK_vs_SHBG_IALGGLLFPASNLR
118 и 18
0,54
0,4442
0,68
0,0027
LBP_ITLPDFTGDLR_vs_C163A_INPASLDK
119 и 54
0,54
0,4604
0,69
0,0013
LBP_ITLPDFTGDLR_vs_CRIS3_AVSPPAR
119 и 78
0,58
0,1395
0,68
0,0035
LBP_ITLPDFTGDLR_vs_CRIS3_YEDLYSNCK
119 и 79
0,59
0,0853
0,67
0,0039
LBP_ITLPDFTGDLR_vs_LYAM1_SYYWIGIR
119 и 120
0,58
0,1601
0,71
6,00E-04
LBP_ITLPDFTGDLR_vs_PGRP2_AGLLRPDYALLGHR
119 и 126
0,56
0,2519
0,69
0,0012
LBP_ITLPDFTGDLR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
119 и 135
0,54
0,4236
0,67
0,0037
LBP_ITLPDFTGDLR_vs_SHBG_IALGGLLFPASNLR
119 и 18
0,56
0,2553
0,7
9,00E-04
LBP_ITLPDFTGDLR_vs_VTDB_ELPEHTVK
119 и 147
0,56
0,3057
0,67
0,0044
PEDF_LQSLFDSPDFSK_vs_CRIS3_AVSPPAR
124 и 78
0,58
0,1461
0,66
0,0074
PEDF_LQSLFDSPDFSK_vs_CRIS3_YEDLYSNCK
124 и 79
0,6
0,082
0,66
0,0088
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,57
0,1856
0,68
0,0027
PEDF_LQSLFDSPDFSK_vs_TENX_LNWEAPPGAFDSFLLR
124 и 141
0,57
0,2324
0,66
0,0065
PEDF_TVQAVLTVPK_vs_CRIS3_YEDLYSNCK
125 и 79
0,57
0,2139
0,66
0,0075
PEDF_TVQAVLTVPK_vs_LYAM1_SYYWIGIR
125 и 120
0,55
0,393
0,69
0,0014
PEDF_TVQAVLTVPK_vs_TENX_LNWEAPPGAFDSFLLR
125 и 141
0,55
0,3856
0,67
0,0045
PSG2_IHPSYTNYR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
133 и 135
0,56
0,2919
0,66
0,0065
VTNC_GQYCYELDEK_vs_IBP3_FLNVLSPR
149 и 99
0,66
0,0043
0,6
0,1065
VTNC_GQYCYELDEK_vs_IBP3_YGQPLPGYTTK
149 и 100
0,69
4,00E-04
0,59
0,1378
VTNC_GQYCYELDEK_vs_IGF2_GIVEECCFR
149 и 103
0,69
4,00E-04
0,6
0,0916
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,65
0,0078
0,65
0,01
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,61
0,0463
0,67
0,0057
VTNC_GQYCYELDEK_vs_PSG3_VSAPSGTGHLPGLNPL
149 и 134
0,7
2,00E-04
0,56
0,3077
VTNC_GQYCYELDEK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
149 и 135
0,57
0,1975
0,66
0,0093
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,62
0,0297
0,67
0,0059
VTNC_GQYCYELDEK_vs_VTDB_ELPEHTVK
149 и 147
0,66
0,0031
0,63
0,0374
VTNC_VDTVDPPYPR_vs_IBP3_YGQPLPGYTTK
150 и 100
0,67
0,0021
0,58
0,1711
VTNC_VDTVDPPYPR_vs_IGF2_GIVEECCFR
150 и 103
0,68
0,0011
0,6
0,1139
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,64
0,0101
0,67
0,0056
VTNC_VDTVDPPYPR_vs_PGRP2_AGLLRPDYALLGHR
150 и 126
0,59
0,0914
0,66
0,0072
VTNC_VDTVDPPYPR_vs_PSG3_VSAPSGTGHLPGLNPL
150 и 134
0,69
4,00E-04
0,55
0,3901
VTNC_VDTVDPPYPR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
150 и 135
0,57
0,2139
0,66
0,0089
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,6
0,0628
0,67
0,0047
VTNC_VDTVDPPYPR_vs_VTDB_ELPEHTVK
150 и 147
0,65
0,0051
0,64
0,0187
Таблица 63: эффективность классификации по обращениям, недели 17-21.
AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, без BMI-стратификации, отдельно для PPROM и PTL.
Обращение
SEQ ID №
119_153_aBMI_35 PTL AUC
119_153_aBMI_35 PTL P-значение
119_153_aBMI_35 PPROM AUC
119_153_aBMI_35 PPROM P-значение
A2GL_DLLLPQPDLR_vs_IBP1_VVESLAK
34 и 97
0,52
0,8126
0,67
0,0107
A2GL_DLLLPQPDLR_vs_LYAM1_SYYWIGIR
34 и 120
0,52
0,8436
0,68
0,0079
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,59
0,3629
0,67
0,0124
AFAM_DADPDTFFAK_vs_LYAM1_SYYWIGIR
37 и 120
0,56
0,5765
0,68
0,0077
AFAM_HFQNLGK_vs_ALS_IRPHTFTGLSGLR
38 и 40
0,54
0,7315
0,66
0,018
AFAM_HFQNLGK_vs_CRIS3_YEDLYSNCK
38 и 79
0,54
0,6701
0,66
0,0228
AFAM_HFQNLGK_vs_IBP1_VVESLAK
38 и 97
0,53
0,7973
0,65
0,0251
AFAM_HFQNLGK_vs_IBP3_FLNVLSPR
38 и 99
0,51
0,959
0,67
0,0126
AFAM_HFQNLGK_vs_IBP3_YGQPLPGYTTK
38 и 100
0,53
0,8024
0,7
0,0037
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,54
0,6798
0,72
0,0014
AFAM_HFQNLGK_vs_LYAM1_SYYWIGIR
38 и 120
0,52
0,8411
0,7
0,0029
AFAM_HFQNLGK_vs_PGRP2_AGLLRPDYALLGHR
38 и 126
0,54
0,7117
0,65
0,025
ANGT_DPTFIPAPIQAK_vs_LYAM1_SYYWIGIR
42 и 120
0,52
0,8101
0,72
0,001
ANGT_DPTFIPAPIQAK_vs_SHBG_IALGGLLFPASNLR
42 и 18
0,53
0,787
0,67
0,0122
APOC3_GWVTDGFSSLK_vs_CRIS3_YEDLYSNCK
47 и 79
0,5
0,9669
0,68
0,0083
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,51
0,9537
0,71
0,0018
APOH_ATVVYQGER_vs_CRIS3_YEDLYSNCK
48 и 79
0,58
0,4588
0,65
0,0262
APOH_ATVVYQGER_vs_IBP1_VVESLAK
48 и 97
0,5
1
0,66
0,0156
APOH_ATVVYQGER_vs_LYAM1_SYYWIGIR
48 и 120
0,57
0,4771
0,68
0,0079
B2MG_VEHSDLSFSK_vs_LYAM1_SYYWIGIR
50 и 120
0,58
0,4409
0,75
2,00E-04
B2MG_VNHVTLSQPK_vs_CRIS3_AVSPPAR
51 и 78
0,53
0,7692
0,68
0,0068
B2MG_VNHVTLSQPK_vs_CRIS3_YEDLYSNCK
51 и 79
0,5
0,9802
0,69
0,0059
B2MG_VNHVTLSQPK_vs_IBP1_VVESLAK
51 и 97
0,55
0,6485
0,68
0,0074
B2MG_VNHVTLSQPK_vs_IGF2_GIVEECCFR
51 и 103
0,51
0,9379
0,68
0,0076
B2MG_VNHVTLSQPK_vs_LYAM1_SYYWIGIR
51 и 120
0,53
0,7516
0,77
1,00E-04
B2MG_VNHVTLSQPK_vs_SHBG_IALGGLLFPASNLR
51 и 18
0,51
0,888
0,68
0,01
CATD_VGFAEAAR_vs_ALS_IRPHTFTGLSGLR
57 и 40
0,51
0,9537
0,69
0,0048
CATD_VGFAEAAR_vs_C163A_INPASLDK
57 и 54
0,51
0,9379
0,77
1,00E-04
CATD_VGFAEAAR_vs_CRIS3_AVSPPAR
57 и 78
0,52
0,8281
0,71
0,0016
CATD_VGFAEAAR_vs_CRIS3_YEDLYSNCK
57 и 79
0,51
0,93
0,72
0,0014
CATD_VGFAEAAR_vs_CSH_AHQLAIDTYQEFEETYIPK
57 и 80
0,57
0,5063
0,7
0,0029
CATD_VGFAEAAR_vs_CSH_ISLLLIESWLEPVR
57 и 81
0,57
0,5063
0,69
0,0064
CATD_VGFAEAAR_vs_FBLN1_TGYYFDGISR
57 и 86
0,54
0,6774
0,71
0,0017
CATD_VGFAEAAR_vs_IBP1_VVESLAK
57 и 97
0,53
0,8024
0,72
0,0012
CATD_VGFAEAAR_vs_IBP2_LIQGAPTIR
57 и 98
0,56
0,5342
0,69
0,0057
CATD_VGFAEAAR_vs_IBP3_FLNVLSPR
57 и 99
0,5
0,9907
0,69
0,0062
CATD_VGFAEAAR_vs_IBP3_YGQPLPGYTTK
57 и 100
0,5
0,9749
0,69
0,0042
CATD_VGFAEAAR_vs_IGF2_GIVEECCFR
57 и 103
0,54
0,6847
0,73
8,00E-04
CATD_VGFAEAAR_vs_ITIH4_ILDDLSPR
57 и 112
0,57
0,5063
0,72
0,0012
CATD_VGFAEAAR_vs_LYAM1_SYYWIGIR
57 и 120
0,52
0,8644
0,78
1,00E-04
CATD_VGFAEAAR_vs_PGRP2_AGLLRPDYALLGHR
57 и 126
0,51
0,9274
0,71
0,0019
CATD_VGFAEAAR_vs_PSG3_VSAPSGTGHLPGLNPL
57 и 134
0,55
0,597
0,75
2,00E-04
CATD_VGFAEAAR_vs_SHBG_IALGGLLFPASNLR
57 и 18
0,5
1
0,71
0,0024
CATD_VGFAEAAR_vs_SOM2.CSH_NYGLLYCFR
57 и 138
0,57
0,4895
0,68
0,0074
CATD_VGFAEAAR_vs_SOM2.CSH_SVEGSCGF
57 и 139
0,61
0,2945
0,68
0,0101
CATD_VGFAEAAR_vs_SPRL1_VLTHSELAPLR
57 и 140
0,51
0,9247
0,72
0,0013
CATD_VGFAEAAR_vs_TENX_LNWEAPPGAFDSFLLR
57 и 141
0,52
0,8462
0,73
7,00E-04
CATD_VGFAEAAR_vs_TENX_LSQLSVTDVTTSSLR
57 и 142
0,53
0,7566
0,72
0,001
CATD_VGFAEAAR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
57 и 144
0,53
0,7845
0,72
0,0014
CATD_VGFAEAAR_vs_VTDB_ELPEHTVK
57 и 147
0,58
0,4311
0,72
0,0011
CATD_VSTLPAITLK_vs_ALS_IRPHTFTGLSGLR
58 и 40
0,52
0,8853
0,69
0,006
CATD_VSTLPAITLK_vs_C163A_INPASLDK
58 и 54
0,5
0,9775
0,73
7,00E-04
CATD_VSTLPAITLK_vs_CRIS3_AVSPPAR
58 и 78
0,5
0,9749
0,71
0,002
CATD_VSTLPAITLK_vs_CRIS3_YEDLYSNCK
58 и 79
0,53
0,7491
0,71
0,0017
CATD_VSTLPAITLK_vs_CSH_AHQLAIDTYQEFEETYIPK
58 и 80
0,57
0,4916
0,68
0,0091
CATD_VSTLPAITLK_vs_CSH_ISLLLIESWLEPVR
58 и 81
0,56
0,5563
0,66
0,0177
CATD_VSTLPAITLK_vs_FBLN1_TGYYFDGISR
58 и 86
0,55
0,639
0,72
0,0013
CATD_VSTLPAITLK_vs_IBP1_VVESLAK
58 и 97
0,52
0,8333
0,72
0,0015
CATD_VSTLPAITLK_vs_IBP3_FLNVLSPR
58 и 99
0,52
0,8436
0,69
0,0049
CATD_VSTLPAITLK_vs_IBP3_YGQPLPGYTTK
58 и 100
0,53
0,7794
0,7
0,0041
CATD_VSTLPAITLK_vs_IGF2_GIVEECCFR
58 и 103
0,57
0,5277
0,72
0,0012
CATD_VSTLPAITLK_vs_ITIH4_ILDDLSPR
58 и 112
0,55
0,6438
0,69
0,0063
CATD_VSTLPAITLK_vs_LYAM1_SYYWIGIR
58 и 120
0,5
0,9907
0,76
1,00E-04
CATD_VSTLPAITLK_vs_PGRP2_AGLLRPDYALLGHR
58 и 126
0,52
0,8359
0,7
0,0027
CATD_VSTLPAITLK_vs_PSG3_VSAPSGTGHLPGLNPL
58 и 134
0,53
0,7415
0,75
3,00E-04
CATD_VSTLPAITLK_vs_SHBG_IALGGLLFPASNLR
58 и 18
0,51
0,8932
0,7
0,003
CATD_VSTLPAITLK_vs_SOM2.CSH_NYGLLYCFR
58 и 138
0,56
0,5342
0,66
0,0194
CATD_VSTLPAITLK_vs_SOM2.CSH_SVEGSCGF
58 и 139
0,6
0,3182
0,65
0,0282
CATD_VSTLPAITLK_vs_SPRL1_VLTHSELAPLR
58 и 140
0,51
0,9116
0,7
0,0038
CATD_VSTLPAITLK_vs_TENX_LNWEAPPGAFDSFLLR
58 и 141
0,55
0,6605
0,72
0,001
CATD_VSTLPAITLK_vs_TENX_LSQLSVTDVTTSSLR
58 и 142
0,51
0,9511
0,71
0,0021
CATD_VSTLPAITLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
58 и 144
0,53
0,7415
0,7
0,0031
CATD_VSTLPAITLK_vs_VTDB_ELPEHTVK
58 и 147
0,55
0,5993
0,7
0,0038
CBPN_EALIQFLEQVHQGIK_vs_LYAM1_SYYWIGIR
59 и 120
0,57
0,5299
0,66
0,0166
CBPN_NGVDLNR_vs_LYAM1_SYYWIGIR
157 и 120
0,59
0,4063
0,68
0,0067
CBPN_NGVDLNR_vs_SPRL1_VLTHSELAPLR
157 и 140
0,56
0,5787
0,65
0,0235
CD14_LTVGAAQVPAQLLVGALR_vs_CRIS3_YEDLYSNCK
61 и 79
0,59
0,3989
0,65
0,0332
CD14_LTVGAAQVPAQLLVGALR_vs_CSH_AHQLAIDTYQEFEETYIPK
61 и 80
0,66
0,1165
0,6
0,1512
CD14_LTVGAAQVPAQLLVGALR_vs_IBP1_VVESLAK
61 и 97
0,52
0,8411
0,68
0,0075
CD14_LTVGAAQVPAQLLVGALR_vs_IGF2_GIVEECCFR
61 и 103
0,62
0,2535
0,66
0,0218
CD14_LTVGAAQVPAQLLVGALR_vs_LYAM1_SYYWIGIR
61 и 120
0,6
0,3208
0,71
0,0016
CD14_LTVGAAQVPAQLLVGALR_vs_PGRP2_AGLLRPDYALLGHR
61 и 126
0,61
0,2972
0,67
0,0121
CD14_LTVGAAQVPAQLLVGALR_vs_SHBG_IALGGLLFPASNLR
61 и 18
0,63
0,2169
0,66
0,0202
CD14_LTVGAAQVPAQLLVGALR_vs_SOM2.CSH_SVEGSCGF
61 и 139
0,68
0,084
0,6
0,1355
CD14_LTVGAAQVPAQLLVGALR_vs_SPRL1_VLTHSELAPLR
61 и 140
0,56
0,5585
0,65
0,0309
CD14_LTVGAAQVPAQLLVGALR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
61 и 144
0,6
0,3508
0,65
0,0271
CD14_LTVGAAQVPAQLLVGALR_vs_VTDB_ELPEHTVK
61 и 147
0,59
0,3734
0,67
0,0112
CD14_SWLAELQQWLKPGLK_vs_CRIS3_YEDLYSNCK
62 и 79
0,56
0,5563
0,65
0,0272
CD14_SWLAELQQWLKPGLK_vs_IBP1_VVESLAK
62 и 97
0,51
0,959
0,69
0,0064
CD14_SWLAELQQWLKPGLK_vs_IGF2_GIVEECCFR
62 и 103
0,57
0,4936
0,66
0,0192
CD14_SWLAELQQWLKPGLK_vs_LYAM1_SYYWIGIR
62 и 120
0,56
0,5765
0,72
0,0013
CD14_SWLAELQQWLKPGLK_vs_PGRP2_AGLLRPDYALLGHR
62 и 126
0,57
0,4916
0,67
0,0113
CD14_SWLAELQQWLKPGLK_vs_SHBG_IALGGLLFPASNLR
62 и 18
0,61
0,3049
0,66
0,0204
CD14_SWLAELQQWLKPGLK_vs_SOM2.CSH_SVEGSCGF
62 и 139
0,65
0,1421
0,61
0,1227
CD14_SWLAELQQWLKPGLK_vs_SPRL1_VLTHSELAPLR
62 и 140
0,53
0,7819
0,65
0,0299
CD14_SWLAELQQWLKPGLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
62 и 144
0,56
0,5652
0,66
0,0169
CD14_SWLAELQQWLKPGLK_vs_VTDB_ELPEHTVK
62 и 147
0,52
0,8749
0,7
0,0042
CFAB_YGLVTYATYPK_vs_LYAM1_SYYWIGIR
64 и 120
0,56
0,5787
0,65
0,0294
CLUS_ASSIIDELFQDR_vs_CRIS3_YEDLYSNCK
67 и 79
0,6
0,3128
0,65
0,0257
CLUS_ASSIIDELFQDR_vs_IBP1_VVESLAK
67 и 97
0,52
0,8671
0,66
0,0187
CLUS_ASSIIDELFQDR_vs_IGF2_GIVEECCFR
67 и 103
0,64
0,1768
0,65
0,0256
CLUS_ASSIIDELFQDR_vs_LYAM1_SYYWIGIR
67 и 120
0,63
0,1944
0,7
0,0028
CLUS_ASSIIDELFQDR_vs_SHBG_IALGGLLFPASNLR
67 и 18
0,62
0,236
0,65
0,0322
CLUS_ASSIIDELFQDR_vs_VTDB_ELPEHTVK
67 и 147
0,65
0,1389
0,64
0,0437
CLUS_LFDSDPITVTVPVEVSR_vs_CRIS3_AVSPPAR
68 и 78
0,57
0,4709
0,65
0,0308
CLUS_LFDSDPITVTVPVEVSR_vs_CRIS3_YEDLYSNCK
68 и 79
0,6
0,3208
0,66
0,0213
CLUS_LFDSDPITVTVPVEVSR_vs_IBP1_VVESLAK
68 и 97
0,52
0,8775
0,66
0,0188
CLUS_LFDSDPITVTVPVEVSR_vs_IGF2_GIVEECCFR
68 и 103
0,65
0,147
0,64
0,0395
CLUS_LFDSDPITVTVPVEVSR_vs_LYAM1_SYYWIGIR
68 и 120
0,62
0,2386
0,69
0,0064
CLUS_LFDSDPITVTVPVEVSR_vs_VTDB_ELPEHTVK
68 и 147
0,63
0,2145
0,65
0,0299
CO5_TLLPVSKPEIR_vs_CRIS3_YEDLYSNCK
70 и 79
0,57
0,5234
0,65
0,0313
CO5_TLLPVSKPEIR_vs_LYAM1_SYYWIGIR
70 и 120
0,6
0,3473
0,7
0,0041
CO5_VFQFLEK_vs_LYAM1_SYYWIGIR
71 и 120
0,59
0,3989
0,67
0,0137
CO6_ALNHLPLEYNSALYSR_vs_LYAM1_SYYWIGIR
72 и 120
0,57
0,4853
0,69
0,0042
CO8A_SLLQPNK_vs_IBP1_VVESLAK
74 и 97
0,5
0,9987
0,66
0,0164
CO8A_SLLQPNK_vs_LYAM1_SYYWIGIR
74 и 120
0,6
0,3525
0,68
0,0076
CO8B_QALEEFQK_vs_IBP1_VVESLAK
76 и 97
0,5
0,9987
0,67
0,012
CO8B_QALEEFQK_vs_LYAM1_SYYWIGIR
76 и 120
0,57
0,5105
0,67
0,0131
F13B_GDTYPAELYITGSILR_vs_CRIS3_AVSPPAR
84 и 78
0,55
0,6179
0,66
0,0157
F13B_GDTYPAELYITGSILR_vs_CRIS3_YEDLYSNCK
84 и 79
0,58
0,4488
0,67
0,0134
F13B_GDTYPAELYITGSILR_vs_IBP1_VVESLAK
84 и 97
0,51
0,9379
0,67
0,0123
F13B_GDTYPAELYITGSILR_vs_IGF2_GIVEECCFR
84 и 103
0,6
0,3273
0,67
0,0146
F13B_GDTYPAELYITGSILR_vs_LYAM1_SYYWIGIR
84 и 120
0,58
0,437
0,74
4,00E-04
F13B_GDTYPAELYITGSILR_vs_SHBG_IALGGLLFPASNLR
84 и 18
0,58
0,4429
0,66
0,0197
F13B_GDTYPAELYITGSILR_vs_SPRL1_VLTHSELAPLR
84 и 140
0,54
0,6677
0,65
0,0254
FETUA_FSVVYAK_vs_CRIS3_YEDLYSNCK
88 и 79
0,55
0,6629
0,69
0,0056
FETUA_FSVVYAK_vs_LYAM1_SYYWIGIR
88 и 120
0,6
0,3456
0,76
1,00E-04
FETUA_HTLNQIDEVK_vs_LYAM1_SYYWIGIR
89 и 120
0,57
0,5212
0,73
7,00E-04
HABP2_FLNWIK_vs_LYAM1_SYYWIGIR
92 и 120
0,6
0,3372
0,72
0,0012
HEMO_NFPSPVDAAFR_vs_LYAM1_SYYWIGIR
93 и 120
0,5
0,996
0,7
0,0041
IBP4_QCHPALDGQR_vs_CRIS3_AVSPPAR
2 и 78
0,53
0,7769
0,71
0,0025
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,52
0,8566
0,71
0,0022
IBP4_QCHPALDGQR_vs_IBP1_VVESLAK
2 и 97
0,54
0,6798
0,69
0,0062
IBP4_QCHPALDGQR_vs_IGF2_GIVEECCFR
2 и 103
0,56
0,5924
0,65
0,0254
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,51
0,9142
0,75
2,00E-04
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,53
0,787
0,67
0,0125
IBP6_HLDSVLQQLQTEVYR_vs_CRIS3_YEDLYSNCK
102 и 79
0,54
0,6994
0,69
0,0063
IBP6_HLDSVLQQLQTEVYR_vs_LYAM1_SYYWIGIR
102 и 120
0,57
0,5277
0,72
0,0016
KNG1_DIPTNSPELEETLTHTITK_vs_IBP1_VVESLAK
116 и 97
0,53
0,7769
0,67
0,0108
KNG1_DIPTNSPELEETLTHTITK_vs_LYAM1_SYYWIGIR
116 и 120
0,52
0,8644
0,7
0,003
KNG1_QVVAGLNFR_vs_CRIS3_YEDLYSNCK
117 и 79
0,6
0,3144
0,65
0,0294
KNG1_QVVAGLNFR_vs_IBP1_VVESLAK
117 и 97
0,51
0,9168
0,68
0,0087
KNG1_QVVAGLNFR_vs_IGF2_GIVEECCFR
117 и 103
0,65
0,1517
0,63
0,0487
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,61
0,2896
0,71
0,0017
KNG1_QVVAGLNFR_vs_SHBG_IALGGLLFPASNLR
117 и 18
0,6
0,3128
0,66
0,0214
LBP_ITLPDFTGDLR_vs_LYAM1_SYYWIGIR
119 и 120
0,55
0,6319
0,71
0,0026
PEDF_LQSLFDSPDFSK_vs_CRIS3_AVSPPAR
124 и 78
0,5
0,9775
0,68
0,0093
PEDF_LQSLFDSPDFSK_vs_CRIS3_YEDLYSNCK
124 и 79
0,52
0,8359
0,68
0,01
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,53
0,787
0,73
9,00E-04
PEDF_TVQAVLTVPK_vs_CRIS3_AVSPPAR
125 и 78
0,5
0,9749
0,68
0,0084
PEDF_TVQAVLTVPK_vs_CRIS3_YEDLYSNCK
125 и 79
0,54
0,729
0,68
0,0071
PEDF_TVQAVLTVPK_vs_IGF2_GIVEECCFR
125 и 103
0,54
0,7216
0,67
0,013
PEDF_TVQAVLTVPK_vs_LYAM1_SYYWIGIR
125 и 120
0,53
0,7491
0,74
4,00E-04
PSG2_IHPSYTNYR_vs_CRIS3_AVSPPAR
133 и 78
0,61
0,2762
0,65
0,0254
PSG2_IHPSYTNYR_vs_CRIS3_YEDLYSNCK
133 и 79
0,63
0,2244
0,66
0,0228
PSG2_IHPSYTNYR_vs_IBP1_VVESLAK
133 и 97
0,58
0,4429
0,65
0,0299
PSG2_IHPSYTNYR_vs_LYAM1_SYYWIGIR
133 и 120
0,63
0,1978
0,67
0,0132
PSG2_IHPSYTNYR_vs_PSG3_VSAPSGTGHLPGLNPL
133 и 134
0,65
0,1498
0,62
0,0668
PSG2_IHPSYTNYR_vs_SHBG_IALGGLLFPASNLR
133 и 18
0,63
0,2194
0,65
0,0315
PTGDS_GPGEDFR_vs_CRIS3_YEDLYSNCK
137 и 79
0,57
0,5105
0,65
0,0303
PTGDS_GPGEDFR_vs_IBP1_VVESLAK
137 и 97
0,51
0,9037
0,67
0,0143
PTGDS_GPGEDFR_vs_LYAM1_SYYWIGIR
137 и 120
0,52
0,8152
0,68
0,0071
THBG_AVLHIGEK_vs_LYAM1_SYYWIGIR
143 и 120
0,56
0,5742
0,68
0,01
VTNC_GQYCYELDEK_vs_CRIS3_YEDLYSNCK
149 и 79
0,56
0,5697
0,66
0,0213
VTNC_GQYCYELDEK_vs_IBP1_VVESLAK
149 и 97
0,51
0,9168
0,67
0,0124
VTNC_GQYCYELDEK_vs_IGF2_GIVEECCFR
149 и 103
0,59
0,3664
0,65
0,0232
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,57
0,5212
0,73
8,00E-04
VTNC_GQYCYELDEK_vs_PSG3_VSAPSGTGHLPGLNPL
149 и 134
0,57
0,5256
0,67
0,0152
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,58
0,4508
0,66
0,0227
VTNC_VDTVDPPYPR_vs_CRIS3_YEDLYSNCK
150 и 79
0,52
0,8307
0,67
0,0145
VTNC_VDTVDPPYPR_vs_IGF2_GIVEECCFR
150 и 103
0,52
0,8307
0,67
0,0143
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,5
0,9669
0,74
4,00E-04
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,53
0,7642
0,66
0,0159
Таблица 64: эффективность классификации по обращениям, недели 17-21.
AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, с BMI-стратификацией (>22≤37), отдельно для PPROM и PTL.
Обращение
SEQ ID №
119_153_rBMI_35 PTL ROC_AUC
119_153_rBMI_35 PTL P-значение
119_153_rBMI_35 PPROM ROC_AUC
119_153_rBMI_35 PPROM P-значение
A2GL_DLLLPQPDLR_vs_CRIS3_YEDLYSNCK
34 и 79
0,59
0,4023
0,67
0,0438
A2GL_DLLLPQPDLR_vs_LYAM1_SYYWIGIR
34 и 120
0,56
0,6023
0,77
0,0014
AFAM_DADPDTFFAK_vs_CRIS3_YEDLYSNCK
37 и 79
0,63
0,2599
0,67
0,0425
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,64
0,2206
0,69
0,0268
AFAM_DADPDTFFAK_vs_LYAM1_SYYWIGIR
37 и 120
0,59
0,4116
0,75
0,0043
AFAM_DADPDTFFAK_vs_PGRP2_AGLLRPDYALLGHR
37 и 126
0,6
0,3694
0,71
0,0154
AFAM_HFQNLGK_vs_ALS_IRPHTFTGLSGLR
38 и 40
0,56
0,6023
0,7
0,0193
AFAM_HFQNLGK_vs_CRIS3_AVSPPAR
38 и 78
0,56
0,5612
0,69
0,0271
AFAM_HFQNLGK_vs_CRIS3_YEDLYSNCK
38 и 79
0,6
0,3665
0,7
0,0184
AFAM_HFQNLGK_vs_IBP3_FLNVLSPR
38 и 99
0,52
0,8893
0,73
0,008
AFAM_HFQNLGK_vs_IBP3_YGQPLPGYTTK
38 и 100
0,53
0,7955
0,73
0,0071
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,58
0,4597
0,75
0,0037
AFAM_HFQNLGK_vs_LYAM1_SYYWIGIR
38 и 120
0,56
0,5686
0,77
0,0014
AFAM_HFQNLGK_vs_PGRP2_AGLLRPDYALLGHR
38 и 126
0,56
0,5797
0,74
0,0051
ANGT_DPTFIPAPIQAK_vs_CRIS3_AVSPPAR
42 и 78
0,52
0,8807
0,73
0,0077
ANGT_DPTFIPAPIQAK_vs_CRIS3_YEDLYSNCK
42 и 79
0,53
0,7913
0,72
0,0088
ANGT_DPTFIPAPIQAK_vs_LYAM1_SYYWIGIR
42 и 120
0,53
0,7704
0,8
5,00E-04
ANGT_DPTFIPAPIQAK_vs_PGRP2_AGLLRPDYALLGHR
42 и 126
0,54
0,7455
0,72
0,0091
ANGT_DPTFIPAPIQAK_vs_SHBG_IALGGLLFPASNLR
42 и 18
0,58
0,4664
0,7
0,02
APOH_ATVVYQGER_vs_LYAM1_SYYWIGIR
48 и 120
0,61
0,3141
0,71
0,0166
APOH_ATVVYQGER_vs_PGRP2_AGLLRPDYALLGHR
48 и 126
0,61
0,3115
0,7
0,0216
B2MG_VEHSDLSFSK_vs_LYAM1_SYYWIGIR
50 и 120
0,53
0,8124
0,79
8,00E-04
B2MG_VNHVTLSQPK_vs_CRIS3_AVSPPAR
51 и 78
0,53
0,7787
0,71
0,0151
B2MG_VNHVTLSQPK_vs_CRIS3_YEDLYSNCK
51 и 79
0,56
0,5723
0,71
0,0151
B2MG_VNHVTLSQPK_vs_LYAM1_SYYWIGIR
51 и 120
0,52
0,8336
0,82
2,00E-04
CATD_VGFAEAAR_vs_ALS_IRPHTFTGLSGLR
57 и 40
0,57
0,5466
0,69
0,024
CATD_VGFAEAAR_vs_C163A_INPASLDK
57 и 54
0,53
0,8039
0,83
1,00E-04
CATD_VGFAEAAR_vs_CRIS3_AVSPPAR
57 и 78
0,54
0,6924
0,74
0,0061
CATD_VGFAEAAR_vs_CRIS3_YEDLYSNCK
57 и 79
0,58
0,4697
0,74
0,0055
CATD_VGFAEAAR_vs_CSH_AHQLAIDTYQEFEETYIPK
57 и 80
0,65
0,1786
0,7
0,0191
CATD_VGFAEAAR_vs_CSH_ISLLLIESWLEPVR
57 и 81
0,65
0,17
0,69
0,0306
CATD_VGFAEAAR_vs_IBP3_FLNVLSPR
57 и 99
0,58
0,5004
0,7
0,0188
CATD_VGFAEAAR_vs_IBP3_YGQPLPGYTTK
57 и 100
0,56
0,5835
0,7
0,0209
CATD_VGFAEAAR_vs_IGF2_GIVEECCFR
57 и 103
0,61
0,3115
0,72
0,0095
CATD_VGFAEAAR_vs_ITIH4_ILDDLSPR
57 и 112
0,52
0,8336
0,74
0,005
CATD_VGFAEAAR_vs_LYAM1_SYYWIGIR
57 и 120
0,55
0,6684
0,82
2,00E-04
CATD_VGFAEAAR_vs_PGRP2_AGLLRPDYALLGHR
57 и 126
0,57
0,5215
0,74
0,0047
CATD_VGFAEAAR_vs_SHBG_IALGGLLFPASNLR
57 и 18
0,56
0,6138
0,7
0,0174
CATD_VGFAEAAR_vs_SOM2.CSH_NYGLLYCFR
57 и 138
0,64
0,2085
0,68
0,0387
CATD_VGFAEAAR_vs_SPRL1_VLTHSELAPLR
57 и 140
0,56
0,6176
0,69
0,0286
CATD_VGFAEAAR_vs_TENX_LNWEAPPGAFDSFLLR
57 и 141
0,53
0,7913
0,73
0,0084
CATD_VSTLPAITLK_vs_ALS_IRPHTFTGLSGLR
58 и 40
0,59
0,4178
0,71
0,0154
CATD_VSTLPAITLK_vs_C163A_INPASLDK
58 и 54
0,52
0,855
0,8
4,00E-04
CATD_VSTLPAITLK_vs_CRIS3_AVSPPAR
58 и 78
0,56
0,5835
0,75
0,0032
CATD_VSTLPAITLK_vs_CRIS3_YEDLYSNCK
58 и 79
0,61
0,3355
0,76
0,0028
CATD_VSTLPAITLK_vs_CSH_AHQLAIDTYQEFEETYIPK
58 и 80
0,65
0,1858
0,7
0,0229
CATD_VSTLPAITLK_vs_CSH_ISLLLIESWLEPVR
58 и 81
0,64
0,2105
0,67
0,0443
CATD_VSTLPAITLK_vs_IBP1_VVESLAK
58 и 97
0,52
0,8507
0,74
0,0056
CATD_VSTLPAITLK_vs_IBP3_FLNVLSPR
58 и 99
0,6
0,3932
0,73
0,0085
CATD_VSTLPAITLK_vs_IBP3_YGQPLPGYTTK
58 и 100
0,59
0,4023
0,71
0,0152
CATD_VSTLPAITLK_vs_IGF2_GIVEECCFR
58 и 103
0,64
0,2186
0,73
0,0065
CATD_VSTLPAITLK_vs_ITIH4_ILDDLSPR
58 и 112
0,5
0,9848
0,73
0,0071
CATD_VSTLPAITLK_vs_LYAM1_SYYWIGIR
58 и 120
0,56
0,5872
0,83
1,00E-04
CATD_VSTLPAITLK_vs_PGRP2_AGLLRPDYALLGHR
58 и 126
0,58
0,463
0,77
0,0017
CATD_VSTLPAITLK_vs_PSG3_VSAPSGTGHLPGLNPL
58 и 134
0,5
0,9674
0,75
0,0042
CATD_VSTLPAITLK_vs_SHBG_IALGGLLFPASNLR
58 и 18
0,57
0,5393
0,72
0,0103
CATD_VSTLPAITLK_vs_TENX_LNWEAPPGAFDSFLLR
58 и 141
0,55
0,6487
0,75
0,0043
CBPN_EALIQFLEQVHQGIK_vs_LYAM1_SYYWIGIR
59 и 120
0,56
0,6099
0,73
0,007
CBPN_NGVDLNR_vs_CRIS3_YEDLYSNCK
157 и 79
0,59
0,4147
0,67
0,0466
CBPN_NGVDLNR_vs_LYAM1_SYYWIGIR
157 и 120
0,59
0,4305
0,75
0,0032
CD14_LTVGAAQVPAQLLVGALR_vs_LYAM1_SYYWIGIR
61 и 120
0,65
0,1858
0,75
0,0038
CD14_LTVGAAQVPAQLLVGALR_vs_PGRP2_AGLLRPDYALLGHR
61 и 126
0,63
0,2289
0,71
0,0156
CD14_LTVGAAQVPAQLLVGALR_vs_SOM2.CSH_SVEGSCGF
61 и 139
0,73
0,0354
0,59
0,315
CD14_SWLAELQQWLKPGLK_vs_LYAM1_SYYWIGIR
62 и 120
0,6
0,3812
0,73
0,0071
CD14_SWLAELQQWLKPGLK_vs_PGRP2_AGLLRPDYALLGHR
62 и 126
0,6
0,3782
0,7
0,0226
CLUS_ASSIIDELFQDR_vs_LYAM1_SYYWIGIR
67 и 120
0,67
0,1206
0,73
0,0083
CLUS_LFDSDPITVTVPVEVSR_vs_LYAM1_SYYWIGIR
68 и 120
0,64
0,2085
0,67
0,0429
CO5_TLLPVSKPEIR_vs_LYAM1_SYYWIGIR
70 и 120
0,65
0,1751
0,72
0,0096
CO5_VFQFLEK_vs_LYAM1_SYYWIGIR
71 и 120
0,64
0,2186
0,68
0,0403
CO6_ALNHLPLEYNSALYSR_vs_LYAM1_SYYWIGIR
72 и 120
0,63
0,2247
0,73
0,0078
CO6_ALNHLPLEYNSALYSR_vs_PGRP2_AGLLRPDYALLGHR
72 и 126
0,64
0,2227
0,67
0,0466
CO8A_SLLQPNK_vs_LYAM1_SYYWIGIR
74 и 120
0,64
0,2027
0,7
0,0204
CO8B_QALEEFQK_vs_LYAM1_SYYWIGIR
76 и 120
0,61
0,341
0,68
0,0412
F13B_GDTYPAELYITGSILR_vs_CRIS3_AVSPPAR
84 и 78
0,62
0,2716
0,69
0,0248
F13B_GDTYPAELYITGSILR_vs_CRIS3_YEDLYSNCK
84 и 79
0,64
0,2027
0,69
0,024
F13B_GDTYPAELYITGSILR_vs_LYAM1_SYYWIGIR
84 и 120
0,62
0,2692
0,79
6,00E-04
F13B_GDTYPAELYITGSILR_vs_PGRP2_AGLLRPDYALLGHR
84 и 126
0,6
0,3812
0,71
0,0166
F13B_GDTYPAELYITGSILR_vs_SHBG_IALGGLLFPASNLR
84 и 18
0,61
0,3194
0,67
0,0456
FETUA_FSVVYAK_vs_LYAM1_SYYWIGIR
88 и 120
0,58
0,4935
0,82
2,00E-04
FETUA_HTLNQIDEVK_vs_LYAM1_SYYWIGIR
89 и 120
0,54
0,7127
0,81
3,00E-04
HEMO_NFPSPVDAAFR_vs_LYAM1_SYYWIGIR
93 и 120
0,53
0,8209
0,73
0,0064
IBP4_QCHPALDGQR_vs_CRIS3_AVSPPAR
2 и 78
0,53
0,7871
0,72
0,0093
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,57
0,5074
0,73
0,007
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,54
0,7372
0,82
2,00E-04
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,58
0,4901
0,68
0,0326
IBP6_HLDSVLQQLQTEVYR_vs_LYAM1_SYYWIGIR
102 и 120
0,53
0,7787
0,78
0,001
ITIH3_ALDLSLK_vs_LYAM1_SYYWIGIR
111 и 120
0,61
0,3328
0,69
0,0302
KNG1_DIPTNSPELEETLTHTITK_vs_LYAM1_SYYWIGIR
116 и 120
0,57
0,525
0,74
0,0045
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,65
0,17
0,75
0,003
PAPP1_DIPHWLNPTR_vs_CRIS3_AVSPPAR
122 и 78
0,62
0,2861
0,7
0,0224
PAPP1_DIPHWLNPTR_vs_CRIS3_YEDLYSNCK
122 и 79
0,65
0,1913
0,69
0,0234
PAPP1_DIPHWLNPTR_vs_LYAM1_SYYWIGIR
122 и 120
0,61
0,3274
0,74
0,006
PAPP1_DIPHWLNPTR_vs_PRG2_WNFAYWAAHQPWSR
122 и 129
0,77
0,0162
0,63
0,1266
PAPP1_DIPHWLNPTR_vs_SHBG_IALGGLLFPASNLR
122 и 18
0,59
0,4369
0,67
0,0434
PAPP1_DIPHWLNPTR_vs_SOM2.CSH_NYGLLYCFR
122 и 138
0,73
0,0405
0,58
0,3298
PAPP1_DIPHWLNPTR_vs_SOM2.CSH_SVEGSCGF
122 и 139
0,77
0,0155
0,65
0,0869
PEDF_LQSLFDSPDFSK_vs_CRIS3_YEDLYSNCK
124 и 79
0,59
0,4336
0,67
0,0466
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,6
0,3522
0,76
0,0029
PEDF_TVQAVLTVPK_vs_CRIS3_AVSPPAR
125 и 78
0,57
0,5109
0,68
0,0319
PEDF_TVQAVLTVPK_vs_CRIS3_YEDLYSNCK
125 и 79
0,6
0,3636
0,69
0,0265
PEDF_TVQAVLTVPK_vs_LYAM1_SYYWIGIR
125 и 120
0,6
0,3551
0,77
0,0016
PEDF_TVQAVLTVPK_vs_PGRP2_AGLLRPDYALLGHR
125 и 126
0,58
0,4498
0,69
0,0302
PSG2_IHPSYTNYR_vs_CRIS3_AVSPPAR
133 и 78
0,67
0,1232
0,72
0,0099
PSG2_IHPSYTNYR_vs_CRIS3_YEDLYSNCK
133 и 79
0,68
0,1011
0,72
0,012
PSG2_IHPSYTNYR_vs_IBP1_VVESLAK
133 и 97
0,59
0,4241
0,67
0,0416
PSG2_IHPSYTNYR_vs_IBP2_LIQGAPTIR
133 и 98
0,67
0,1232
0,69
0,0268
PSG2_IHPSYTNYR_vs_IGF2_GIVEECCFR
133 и 103
0,66
0,1429
0,67
0,0429
PSG2_IHPSYTNYR_vs_LYAM1_SYYWIGIR
133 и 120
0,68
0,0966
0,76
0,0024
PSG2_IHPSYTNYR_vs_PGRP2_AGLLRPDYALLGHR
133 и 126
0,66
0,1414
0,7
0,0204
PSG2_IHPSYTNYR_vs_PSG3_VSAPSGTGHLPGLNPL
133 и 134
0,68
0,1081
0,67
0,0456
PSG2_IHPSYTNYR_vs_SHBG_IALGGLLFPASNLR
133 и 18
0,68
0,1057
0,71
0,0134
PSG2_IHPSYTNYR_vs_SOM2.CSH_SVEGSCGF
133 и 139
0,74
0,0336
0,64
0,1069
PTGDS_GPGEDFR_vs_LYAM1_SYYWIGIR
137 и 120
0,6
0,3812
0,72
0,0088
THBG_AVLHIGEK_vs_LYAM1_SYYWIGIR
143 и 120
0,61
0,341
0,69
0,0265
VTNC_GQYCYELDEK_vs_CRIS3_AVSPPAR
149 и 78
0,6
0,3871
0,68
0,0383
VTNC_GQYCYELDEK_vs_CRIS3_YEDLYSNCK
149 и 79
0,62
0,2886
0,69
0,0245
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,62
0,2788
0,79
8,00E-04
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,62
0,2986
0,7
0,0209
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,61
0,3355
0,67
0,0476
VTNC_VDTVDPPYPR_vs_CRIS3_YEDLYSNCK
150 и 79
0,57
0,5144
0,69
0,0286
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,55
0,6253
0,78
0,0012
Таблица 65: эффективность классификации по обращениям, недели 17-21.
AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, без BMI-стратификации, отдельно для первой беременности и повторной беременности.
Обращение
SEQ ID №
119_153_aBMI_37 multi ROC_AUC
119_153_aBMI_37 multi P-значение
119_153_aBMI_37 primi ROC_AUC
119_153_aBMI_37 primi P-значение
A2GL_DLLLPQPDLR_vs_SHBG_IALGGLLFPASNLR
34 и 18
0,59
0,0399
0,67
0,0102
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,65
4,00E-04
0,53
0,6818
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,65
5,00E-04
0,56
0,3992
APOC3_GWVTDGFSSLK_vs_IGF2_GIVEECCFR
47 и 103
0,65
3,00E-04
0,62
0,0687
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,63
0,0017
0,65
0,0238
APOC3_GWVTDGFSSLK_vs_PGRP2_AGLLRPDYALLGHR
47 и 126
0,62
0,0049
0,67
0,0104
APOC3_GWVTDGFSSLK_vs_PSG3_VSAPSGTGHLPGLNPL
47 и 134
0,65
4,00E-04
0,65
0,0277
APOC3_GWVTDGFSSLK_vs_SHBG_IALGGLLFPASNLR
47 и 18
0,62
0,0049
0,66
0,0148
APOC3_GWVTDGFSSLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
47 и 144
0,62
0,0036
0,65
0,0218
APOH_ATVVYQGER_vs_SHBG_IALGGLLFPASNLR
48 и 18
0,59
0,0394
0,65
0,0255
B2MG_VNHVTLSQPK_vs_SHBG_IALGGLLFPASNLR
51 и 18
0,57
0,0849
0,7
0,0029
C1QB_VPGLYYFTYHASSR_vs_PGRP2_AGLLRPDYALLGHR
55 и 126
0,57
0,1204
0,68
0,0058
C1QB_VPGLYYFTYHASSR_vs_PSG3_VSAPSGTGHLPGLNPL
55 и 134
0,58
0,0476
0,66
0,0171
C1QB_VPGLYYFTYHASSR_vs_SHBG_IALGGLLFPASNLR
55 и 18
0,58
0,0754
0,71
0,0017
C1QB_VPGLYYFTYHASSR_vs_TENX_LNWEAPPGAFDSFLLR
55 и 141
0,58
0,0678
0,68
0,0077
CATD_VGFAEAAR_vs_SHBG_IALGGLLFPASNLR
57 и 18
0,57
0,0986
0,68
0,0055
CATD_VGFAEAAR_vs_TENX_LNWEAPPGAFDSFLLR
57 и 141
0,58
0,0743
0,69
0,0038
CATD_VSTLPAITLK_vs_SHBG_IALGGLLFPASNLR
58 и 18
0,57
0,1127
0,69
0,0049
CATD_VSTLPAITLK_vs_TENX_LNWEAPPGAFDSFLLR
58 и 141
0,57
0,1066
0,69
0,0035
CD14_LTVGAAQVPAQLLVGALR_vs_SHBG_IALGGLLFPASNLR
61 и 18
0,58
0,0737
0,68
0,0068
CD14_LTVGAAQVPAQLLVGALR_vs_TENX_LNWEAPPGAFDSFLLR
61 и 141
0,58
0,0622
0,66
0,0135
CD14_SWLAELQQWLKPGLK_vs_SHBG_IALGGLLFPASNLR
62 и 18
0,59
0,0371
0,66
0,0148
CD14_SWLAELQQWLKPGLK_vs_TENX_LNWEAPPGAFDSFLLR
62 и 141
0,59
0,0404
0,65
0,023
CFAB_YGLVTYATYPK_vs_IGF2_GIVEECCFR
64 и 103
0,66
2,00E-04
0,55
0,493
CFAB_YGLVTYATYPK_vs_PGRP2_AGLLRPDYALLGHR
64 и 126
0,6
0,0247
0,65
0,0255
CFAB_YGLVTYATYPK_vs_PSG3_VSAPSGTGHLPGLNPL
64 и 134
0,65
6,00E-04
0,61
0,1087
CO5_TLLPVSKPEIR_vs_SHBG_IALGGLLFPASNLR
70 и 18
0,58
0,066
0,69
0,0049
CO5_TLLPVSKPEIR_vs_TENX_LNWEAPPGAFDSFLLR
70 и 141
0,58
0,0557
0,67
0,0119
CO5_VFQFLEK_vs_SHBG_IALGGLLFPASNLR
71 и 18
0,59
0,0314
0,66
0,013
CO6_ALNHLPLEYNSALYSR_vs_SHBG_IALGGLLFPASNLR
72 и 18
0,58
0,0515
0,65
0,0234
CO8A_SLLQPNK_vs_IGF2_GIVEECCFR
74 и 103
0,66
2,00E-04
0,52
0,8158
CO8B_QALEEFQK_vs_IGF2_GIVEECCFR
76 и 103
0,66
2,00E-04
0,53
0,6297
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_SHBG_IALGGLLFPASNLR
82 и 18
0,61
0,0121
0,66
0,0171
ENPP2_TYLHTYESEI_vs_PSG3_VSAPSGTGHLPGLNPL
83 и 134
0,59
0,0287
0,65
0,023
ENPP2_TYLHTYESEI_vs_SHBG_IALGGLLFPASNLR
83 и 18
0,59
0,0253
0,66
0,0135
FBLN3_IPSNPSHR_vs_SHBG_IALGGLLFPASNLR
87 и 18
0,55
0,2452
0,73
4,00E-04
FETUA_FSVVYAK_vs_IGF2_GIVEECCFR
88 и 103
0,68
0
0,51
0,862
FETUA_HTLNQIDEVK_vs_IGF2_GIVEECCFR
89 и 103
0,66
2,00E-04
0,51
0,9138
HABP2_FLNWIK_vs_SHBG_IALGGLLFPASNLR
92 и 18
0,59
0,0407
0,66
0,0162
HABP2_FLNWIK_vs_TENX_LNWEAPPGAFDSFLLR
92 и 141
0,59
0,0277
0,67
0,0108
HEMO_NFPSPVDAAFR_vs_SHBG_IALGGLLFPASNLR
93 и 18
0,59
0,0432
0,67
0,0088
HEMO_NFPSPVDAAFR_vs_TENX_LNWEAPPGAFDSFLLR
93 и 141
0,58
0,0642
0,66
0,0128
IBP4_QCHPALDGQR_vs_PGRP2_AGLLRPDYALLGHR
2 и 126
0,57
0,0774
0,73
5,00E-04
IBP4_QCHPALDGQR_vs_PSG3_VSAPSGTGHLPGLNPL
2 и 134
0,63
0,0017
0,67
0,0095
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,6
0,0241
0,72
7,00E-04
IBP4_QCHPALDGQR_vs_TENX_LNWEAPPGAFDSFLLR
2 и 141
0,59
0,0444
0,69
0,0048
INHBC_LDFHFSSDR_vs_IBP3_YGQPLPGYTTK
107 и 100
0,65
6,00E-04
0,54
0,5224
INHBC_LDFHFSSDR_vs_IGF2_GIVEECCFR
107 и 103
0,67
1,00E-04
0,55
0,4254
INHBC_LDFHFSSDR_vs_PGRP2_AGLLRPDYALLGHR
107 и 126
0,61
0,009
0,65
0,0242
INHBC_LDFHFSSDR_vs_PSG3_VSAPSGTGHLPGLNPL
107 и 134
0,66
2,00E-04
0,59
0,1755
ITIH3_ALDLSLK_vs_SHBG_IALGGLLFPASNLR
111 и 18
0,61
0,0073
0,65
0,0207
KNG1_DIPTNSPELEETLTHTITK_vs_PSG3_VSAPSGTGHLPGLNPL
116 и 134
0,59
0,0285
0,66
0,0138
KNG1_DIPTNSPELEETLTHTITK_vs_SHBG_IALGGLLFPASNLR
116 и 18
0,56
0,1468
0,69
0,0045
KNG1_QVVAGLNFR_vs_SHBG_IALGGLLFPASNLR
117 и 18
0,56
0,1453
0,68
0,0062
LBP_ITGFLKPGK_vs_PSG3_VSAPSGTGHLPGLNPL
118 и 134
0,65
3,00E-04
0,61
0,0989
LBP_ITGFLKPGK_vs_SHBG_IALGGLLFPASNLR
118 и 18
0,61
0,0097
0,65
0,0277
LBP_ITLPDFTGDLR_vs_CRIS3_AVSPPAR
119 и 78
0,65
5,00E-04
0,55
0,4254
LBP_ITLPDFTGDLR_vs_CRIS3_YEDLYSNCK
119 и 79
0,65
3,00E-04
0,57
0,3108
LBP_ITLPDFTGDLR_vs_IGF2_GIVEECCFR
119 и 103
0,65
4,00E-04
0,57
0,3015
LBP_ITLPDFTGDLR_vs_PGRP2_AGLLRPDYALLGHR
119 и 126
0,61
0,0103
0,67
0,0116
LBP_ITLPDFTGDLR_vs_PSG3_VSAPSGTGHLPGLNPL
119 и 134
0,66
1,00E-04
0,64
0,0397
LBP_ITLPDFTGDLR_vs_SHBG_IALGGLLFPASNLR
119 и 18
0,62
0,0039
0,66
0,0128
LBP_ITLPDFTGDLR_vs_TENX_LNWEAPPGAFDSFLLR
119 и 141
0,61
0,0132
0,65
0,0218
PEDF_TVQAVLTVPK_vs_TENX_LNWEAPPGAFDSFLLR
125 и 141
0,59
0,0331
0,65
0,0222
VTNC_GQYCYELDEK_vs_IGF2_GIVEECCFR
149 и 103
0,66
1,00E-04
0,57
0,2689
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,59
0,0262
0,68
0,0069
VTNC_GQYCYELDEK_vs_PSG3_VSAPSGTGHLPGLNPL
149 и 134
0,65
6,00E-04
0,65
0,0218
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,62
0,0039
0,68
0,0058
VTNC_GQYCYELDEK_vs_TENX_LNWEAPPGAFDSFLLR
149 и 141
0,6
0,0134
0,65
0,0204
VTNC_GQYCYELDEK_vs_VTDB_ELPEHTVK
149 и 147
0,62
0,0036
0,65
0,0255
VTNC_VDTVDPPYPR_vs_IGF2_GIVEECCFR
150 и 103
0,66
1,00E-04
0,56
0,3883
VTNC_VDTVDPPYPR_vs_PGRP2_AGLLRPDYALLGHR
150 и 126
0,59
0,0402
0,68
0,0078
VTNC_VDTVDPPYPR_vs_PSG3_VSAPSGTGHLPGLNPL
150 и 134
0,64
7,00E-04
0,65
0,0215
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,61
0,0068
0,67
0,0114
VTNC_VDTVDPPYPR_vs_VTDB_ELPEHTVK
150 и 147
0,65
4,00E-04
0,63
0,0558
Таблица 66: эффективность классификации по обращениям, недели 17-21.
AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, с BMI-стратификацией (>22≤37), отдельно для первой беременности и повторной беременности.
Обращение
SEQ ID №
119_153_rBMI_37 multi ROC_AUC
119_153_rBMI_37 multi P-значение
119_153_rBMI_37 primi ROC_AUC
119_153_rBMI_37 primi P-значение
A2GL_DLLLPQPDLR_vs_CRIS3_AVSPPAR
34 и 78
0,65
0,0038
0,52
0,8315
A2GL_DLLLPQPDLR_vs_CRIS3_YEDLYSNCK
34 и 79
0,65
0,0037
0,52
0,8126
A2GL_DLLLPQPDLR_vs_IGF2_GIVEECCFR
34 и 103
0,65
0,0026
0,51
0,9467
A2GL_DLLLPQPDLR_vs_LYAM1_SYYWIGIR
34 и 120
0,62
0,0209
0,65
0,071
A2GL_DLLLPQPDLR_vs_PGRP2_AGLLRPDYALLGHR
34 и 126
0,57
0,1962
0,72
0,0073
A2GL_DLLLPQPDLR_vs_SHBG_IALGGLLFPASNLR
34 и 18
0,59
0,0826
0,69
0,0219
AFAM_DADPDTFFAK_vs_IBP3_FLNVLSPR
37 и 99
0,67
9,00E-04
0,57
0,4118
AFAM_DADPDTFFAK_vs_IBP3_YGQPLPGYTTK
37 и 100
0,68
3,00E-04
0,55
0,5885
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,71
0
0,57
0,4118
AFAM_HFQNLGK_vs_IBP3_FLNVLSPR
38 и 99
0,67
9,00E-04
0,51
0,8792
AFAM_HFQNLGK_vs_IBP3_YGQPLPGYTTK
38 и 100
0,68
4,00E-04
0,5
0,9854
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,7
1,00E-04
0,53
0,7565
ANGT_DPTFIPAPIQAK_vs_CRIS3_AVSPPAR
42 и 78
0,66
0,001
0,54
0,6484
ANGT_DPTFIPAPIQAK_vs_CRIS3_YEDLYSNCK
42 и 79
0,66
0,0016
0,5
1
ANGT_DPTFIPAPIQAK_vs_IGF2_GIVEECCFR
42 и 103
0,65
0,003
0,51
0,8696
ANGT_DPTFIPAPIQAK_vs_SHBG_IALGGLLFPASNLR
42 и 18
0,6
0,0486
0,69
0,0212
ANGT_DPTFIPAPIQAK_vs_TENX_LNWEAPPGAFDSFLLR
42 и 141
0,57
0,1908
0,71
0,0121
APOC3_GWVTDGFSSLK_vs_C163A_INPASLDK
47 и 54
0,6
0,0479
0,68
0,0291
APOC3_GWVTDGFSSLK_vs_CRIS3_YEDLYSNCK
47 и 79
0,63
0,0104
0,65
0,0673
APOC3_GWVTDGFSSLK_vs_IBP3_FLNVLSPR
47 и 99
0,61
0,0229
0,65
0,0789
APOC3_GWVTDGFSSLK_vs_IBP3_YGQPLPGYTTK
47 и 100
0,61
0,036
0,65
0,0789
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,6
0,0418
0,71
0,0129
APOC3_GWVTDGFSSLK_vs_PGRP2_AGLLRPDYALLGHR
47 и 126
0,57
0,1362
0,72
0,0088
APOC3_GWVTDGFSSLK_vs_PSG3_VSAPSGTGHLPGLNPL
47 и 134
0,6
0,0572
0,69
0,0257
APOC3_GWVTDGFSSLK_vs_SHBG_IALGGLLFPASNLR
47 и 18
0,58
0,1164
0,7
0,0186
APOC3_GWVTDGFSSLK_vs_TENX_LNWEAPPGAFDSFLLR
47 и 141
0,58
0,1194
0,7
0,0163
APOC3_GWVTDGFSSLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
47 и 144
0,58
0,0932
0,67
0,0393
APOC3_GWVTDGFSSLK_vs_VTDB_ELPEHTVK
47 и 147
0,58
0,1312
0,69
0,0249
APOH_ATVVYQGER_vs_IBP3_YGQPLPGYTTK
48 и 100
0,65
0,0021
0,51
0,908
APOH_ATVVYQGER_vs_IGF2_GIVEECCFR
48 и 103
0,67
5,00E-04
0,58
0,3523
B2MG_VNHVTLSQPK_vs_IGF2_GIVEECCFR
51 и 103
0,65
0,0034
0,5
1
B2MG_VNHVTLSQPK_vs_LYAM1_SYYWIGIR
51 и 120
0,57
0,1547
0,68
0,0339
BGH3_LTLLAPLNSVFK_vs_CRIS3_AVSPPAR
52 и 78
0,65
0,0022
0,52
0,841
BGH3_LTLLAPLNSVFK_vs_CRIS3_YEDLYSNCK
52 и 79
0,66
0,0015
0,5
0,9854
BGH3_LTLLAPLNSVFK_vs_IGF2_GIVEECCFR
52 и 103
0,66
0,0014
0,56
0,4998
BGH3_LTLLAPLNSVFK_vs_TENX_LNWEAPPGAFDSFLLR
52 и 141
0,58
0,112
0,66
0,0496
C1QB_VPGLYYFTYHASSR_vs_PGRP2_AGLLRPDYALLGHR
55 и 126
0,57
0,1483
0,71
0,0109
C1QB_VPGLYYFTYHASSR_vs_SHBG_IALGGLLFPASNLR
55 и 18
0,57
0,1962
0,72
0,0073
C1QB_VPGLYYFTYHASSR_vs_TENX_LNWEAPPGAFDSFLLR
55 и 141
0,55
0,3241
0,72
0,0082
CBPN_NGVDLNR_vs_IGF2_GIVEECCFR
157 и 103
0,66
0,0017
0,55
0,5392
CD14_LTVGAAQVPAQLLVGALR_vs_IGF2_GIVEECCFR
61 и 103
0,65
0,0038
0,51
0,8696
CFAB_YGLVTYATYPK_vs_CRIS3_AVSPPAR
64 и 78
0,65
0,0037
0,53
0,6838
CFAB_YGLVTYATYPK_vs_CRIS3_YEDLYSNCK
64 и 79
0,65
0,0033
0,5
0,9951
CFAB_YGLVTYATYPK_vs_IGF2_GIVEECCFR
64 и 103
0,67
8,00E-04
0,51
0,9273
CFAB_YGLVTYATYPK_vs_PGRP2_AGLLRPDYALLGHR
64 и 126
0,57
0,1865
0,7
0,0175
CFAB_YGLVTYATYPK_vs_TENX_LNWEAPPGAFDSFLLR
64 и 141
0,55
0,3241
0,7
0,018
CLUS_ASSIIDELFQDR_vs_IGF2_GIVEECCFR
67 и 103
0,65
0,0021
0,54
0,6311
CO5_TLLPVSKPEIR_vs_IGF2_GIVEECCFR
70 и 103
0,65
0,0028
0,51
0,9177
CO5_TLLPVSKPEIR_vs_LYAM1_SYYWIGIR
70 и 120
0,6
0,0461
0,68
0,0291
CO5_TLLPVSKPEIR_vs_PGRP2_AGLLRPDYALLGHR
70 и 126
0,55
0,2997
0,74
0,0035
CO5_TLLPVSKPEIR_vs_PSG3_VSAPSGTGHLPGLNPL
70 и 134
0,59
0,0814
0,68
0,0309
CO5_TLLPVSKPEIR_vs_SHBG_IALGGLLFPASNLR
70 и 18
0,57
0,1519
0,73
0,0066
CO5_TLLPVSKPEIR_vs_TENX_LNWEAPPGAFDSFLLR
70 и 141
0,54
0,4195
0,75
0,0032
CO5_VFQFLEK_vs_CRIS3_AVSPPAR
71 и 78
0,65
0,0033
0,55
0,5801
CO5_VFQFLEK_vs_IGF2_GIVEECCFR
71 и 103
0,65
0,0035
0,54
0,6397
CO5_VFQFLEK_vs_PGRP2_AGLLRPDYALLGHR
71 и 126
0,55
0,2779
0,73
0,0068
CO5_VFQFLEK_vs_SHBG_IALGGLLFPASNLR
71 и 18
0,58
0,1217
0,69
0,0199
CO5_VFQFLEK_vs_TENX_LNWEAPPGAFDSFLLR
71 и 141
0,55
0,3581
0,72
0,0076
CO6_ALNHLPLEYNSALYSR_vs_IGF2_GIVEECCFR
72 и 103
0,66
0,0016
0,54
0,6311
CO6_ALNHLPLEYNSALYSR_vs_PGRP2_AGLLRPDYALLGHR
72 и 126
0,56
0,2613
0,7
0,0148
CO8A_SLLQPNK_vs_IGF2_GIVEECCFR
74 и 103
0,66
0,0015
0,52
0,7751
CO8A_SLLQPNK_vs_TENX_LNWEAPPGAFDSFLLR
74 и 141
0,55
0,2952
0,72
0,0094
CO8B_QALEEFQK_vs_IGF2_GIVEECCFR
76 и 103
0,66
0,0018
0,51
0,9273
CO8B_QALEEFQK_vs_TENX_LNWEAPPGAFDSFLLR
76 и 141
0,55
0,2894
0,72
0,0076
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_SHBG_IALGGLLFPASNLR
82 и 18
0,6
0,058
0,69
0,0206
ENPP2_TYLHTYESEI_vs_LYAM1_SYYWIGIR
83 и 120
0,57
0,1612
0,67
0,0429
ENPP2_TYLHTYESEI_vs_SHBG_IALGGLLFPASNLR
83 и 18
0,56
0,1973
0,7
0,0175
F13B_GDTYPAELYITGSILR_vs_IGF2_GIVEECCFR
84 и 103
0,65
0,0029
0,52
0,8315
FETUA_FSVVYAK_vs_IGF2_GIVEECCFR
88 и 103
0,69
1,00E-04
0,57
0,4188
FETUA_HTLNQIDEVK_vs_IGF2_GIVEECCFR
89 и 103
0,68
4,00E-04
0,57
0,3847
HABP2_FLNWIK_vs_IGF2_GIVEECCFR
92 и 103
0,65
0,0025
0,58
0,3158
HABP2_FLNWIK_vs_TENX_LNWEAPPGAFDSFLLR
92 и 141
0,54
0,4049
0,74
0,0033
IBP4_QCHPALDGQR_vs_CRIS3_AVSPPAR
2 и 78
0,65
0,0034
0,54
0,6224
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,65
0,0022
0,57
0,3847
IBP4_QCHPALDGQR_vs_IGF2_GIVEECCFR
2 и 103
0,68
3,00E-04
0,54
0,6139
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,62
0,022
0,7
0,0163
IBP4_QCHPALDGQR_vs_PGRP2_AGLLRPDYALLGHR
2 и 126
0,58
0,1353
0,77
0,0013
IBP4_QCHPALDGQR_vs_PSG3_VSAPSGTGHLPGLNPL
2 и 134
0,59
0,0656
0,69
0,0199
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,59
0,0724
0,75
0,0027
IBP4_QCHPALDGQR_vs_TENX_LNWEAPPGAFDSFLLR
2 и 141
0,54
0,3716
0,75
0,0025
IBP4_QCHPALDGQR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
2 и 144
0,59
0,0826
0,67
0,0455
IBP4_QCHPALDGQR_vs_VTDB_ELPEHTVK
2 и 147
0,57
0,17
0,7
0,0158
INHBC_LDFHFSSDR_vs_ALS_IRPHTFTGLSGLR
107 и 40
0,65
0,0036
0,53
0,7658
INHBC_LDFHFSSDR_vs_CRIS3_YEDLYSNCK
107 и 79
0,65
0,0033
0,52
0,8601
INHBC_LDFHFSSDR_vs_IBP3_FLNVLSPR
107 и 99
0,66
0,0011
0,52
0,8505
INHBC_LDFHFSSDR_vs_IBP3_YGQPLPGYTTK
107 и 100
0,67
9,00E-04
0,53
0,7381
INHBC_LDFHFSSDR_vs_IGF2_GIVEECCFR
107 и 103
0,69
2,00E-04
0,51
0,8984
INHBC_LDFHFSSDR_vs_PGRP2_AGLLRPDYALLGHR
107 и 126
0,59
0,0661
0,67
0,036
INHBC_LDFHFSSDR_vs_SHBG_IALGGLLFPASNLR
107 и 18
0,6
0,0414
0,65
0,0655
INHBC_LDFHFSSDR_vs_TENX_LNWEAPPGAFDSFLLR
107 и 141
0,6
0,0447
0,73
0,0068
INHBC_LDFHFSSDR_vs_TENX_LSQLSVTDVTTSSLR
107 и 142
0,59
0,075
0,68
0,0319
INHBC_LDFHFSSDR_vs_VTDB_ELPEHTVK
107 и 147
0,62
0,0177
0,65
0,081
ITIH3_ALDLSLK_vs_CRIS3_AVSPPAR
111 и 78
0,66
0,002
0,54
0,6572
ITIH3_ALDLSLK_vs_CRIS3_YEDLYSNCK
111 и 79
0,65
0,0031
0,51
0,937
ITIH3_ALDLSLK_vs_SHBG_IALGGLLFPASNLR
111 и 18
0,61
0,031
0,65
0,0673
KNG1_DIPTNSPELEETLTHTITK_vs_LYAM1_SYYWIGIR
116 и 120
0,57
0,1519
0,67
0,0417
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,57
0,1632
0,68
0,0349
LBP_ITLPDFTGDLR_vs_CRIS3_AVSPPAR
119 и 78
0,66
0,0016
0,53
0,7381
LBP_ITLPDFTGDLR_vs_CRIS3_YEDLYSNCK
119 и 79
0,66
0,002
0,57
0,4188
LBP_ITLPDFTGDLR_vs_IGF2_GIVEECCFR
119 и 103
0,66
0,0018
0,5
0,9854
LBP_ITLPDFTGDLR_vs_PGRP2_AGLLRPDYALLGHR
119 и 126
0,59
0,0661
0,69
0,0219
LBP_ITLPDFTGDLR_vs_SHBG_IALGGLLFPASNLR
119 и 18
0,61
0,0352
0,66
0,0525
LBP_ITLPDFTGDLR_vs_TENX_LNWEAPPGAFDSFLLR
119 и 141
0,56
0,2302
0,68
0,0291
PEDF_LQSLFDSPDFSK_vs_CRIS3_AVSPPAR
124 и 78
0,67
6,00E-04
0,54
0,6749
PEDF_LQSLFDSPDFSK_vs_CRIS3_YEDLYSNCK
124 и 79
0,67
0,001
0,5
0,9661
PEDF_LQSLFDSPDFSK_vs_TENX_LNWEAPPGAFDSFLLR
124 и 141
0,56
0,2086
0,71
0,0109
PEDF_TVQAVLTVPK_vs_IGF2_GIVEECCFR
125 и 103
0,66
0,0019
0,55
0,5312
PEDF_TVQAVLTVPK_vs_TENX_LNWEAPPGAFDSFLLR
125 и 141
0,54
0,3854
0,73
0,0055
PSG2_IHPSYTNYR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
133 и 135
0,67
5,00E-04
0,58
0,3399
VTNC_GQYCYELDEK_vs_CRIS3_AVSPPAR
149 и 78
0,66
0,0014
0,51
0,9177
VTNC_GQYCYELDEK_vs_CRIS3_YEDLYSNCK
149 и 79
0,66
0,0018
0,55
0,5718
VTNC_GQYCYELDEK_vs_IBP3_FLNVLSPR
149 и 99
0,66
0,002
0,56
0,4845
VTNC_GQYCYELDEK_vs_IBP3_YGQPLPGYTTK
149 и 100
0,68
4,00E-04
0,56
0,4921
VTNC_GQYCYELDEK_vs_IGF2_GIVEECCFR
149 и 103
0,7
1,00E-04
0,51
0,9273
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,64
0,0058
0,67
0,0442
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,59
0,061
0,72
0,0076
VTNC_GQYCYELDEK_vs_PSG3_VSAPSGTGHLPGLNPL
149 и 134
0,62
0,0184
0,68
0,0273
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,62
0,0198
0,71
0,0125
VTNC_GQYCYELDEK_vs_TENX_LNWEAPPGAFDSFLLR
149 и 141
0,58
0,1345
0,72
0,0085
VTNC_GQYCYELDEK_vs_VTDB_ELPEHTVK
149 и 147
0,63
0,0085
0,68
0,0291
VTNC_VDTVDPPYPR_vs_CRIS3_AVSPPAR
150 и 78
0,66
0,0014
0,5
0,9661
VTNC_VDTVDPPYPR_vs_CRIS3_YEDLYSNCK
150 и 79
0,66
0,0018
0,54
0,6484
VTNC_VDTVDPPYPR_vs_IBP3_YGQPLPGYTTK
150 и 100
0,66
0,002
0,56
0,4998
VTNC_VDTVDPPYPR_vs_IGF2_GIVEECCFR
150 и 103
0,68
3,00E-04
0,5
0,9854
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,64
0,0066
0,69
0,0249
VTNC_VDTVDPPYPR_vs_PGRP2_AGLLRPDYALLGHR
150 и 126
0,58
0,1345
0,72
0,0079
VTNC_VDTVDPPYPR_vs_PSG3_VSAPSGTGHLPGLNPL
150 и 134
0,6
0,0516
0,72
0,0098
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,6
0,0375
0,7
0,0175
VTNC_VDTVDPPYPR_vs_TENX_LNWEAPPGAFDSFLLR
150 и 141
0,57
0,1962
0,71
0,0101
VTNC_VDTVDPPYPR_vs_VTDB_ELPEHTVK
150 и 147
0,64
0,0066
0,68
0,0339
Таблица 67: эффективность классификации по обращениям, недели 17-21.
AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, без BMI-стратификации, отдельно для первой беременности и повторной беременности.
Обращение
SEQ ID №
119_153_aBMI_35 multi ROC_AUC
119_153_aBMI_35 multi P-значение
119_153_aBMI_35 primi ROC_AUC
119_153_aBMI_35 primi P-значение
A2GL_DLLLPQPDLR_vs_IBP1_VVESLAK
34 и 97
0,65
0,0463
0,58
0,4028
A2GL_DLLLPQPDLR_vs_LYAM1_SYYWIGIR
34 и 120
0,68
0,0136
0,61
0,2481
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,7
0,0065
0,56
0,5483
AFAM_DADPDTFFAK_vs_PGRP2_AGLLRPDYALLGHR
37 и 126
0,55
0,5449
0,73
0,0121
AFAM_DADPDTFFAK_vs_SHBG_IALGGLLFPASNLR
37 и 18
0,54
0,6027
0,71
0,0204
AFAM_HFQNLGK_vs_ALS_IRPHTFTGLSGLR
38 и 40
0,69
0,0121
0,5
1
AFAM_HFQNLGK_vs_CRIS3_YEDLYSNCK
38 и 79
0,69
0,0122
0,54
0,6691
AFAM_HFQNLGK_vs_IBP3_FLNVLSPR
38 и 99
0,69
0,0097
0,51
0,9348
AFAM_HFQNLGK_vs_IBP3_YGQPLPGYTTK
38 и 100
0,74
0,0012
0,52
0,8343
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,73
0,0021
0,55
0,5667
AFAM_HFQNLGK_vs_PGRP2_AGLLRPDYALLGHR
38 и 126
0,55
0,5034
0,73
0,0137
ANGT_DPTFIPAPIQAK_vs_LYAM1_SYYWIGIR
42 и 120
0,72
0,0028
0,59
0,335
ANGT_DPTFIPAPIQAK_vs_SHBG_IALGGLLFPASNLR
42 и 18
0,58
0,2847
0,71
0,0219
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,61
0,1507
0,75
0,0069
APOH_ATVVYQGER_vs_CRIS3_YEDLYSNCK
48 и 79
0,71
0,0043
0,53
0,778
APOH_ATVVYQGER_vs_LYAM1_SYYWIGIR
48 и 120
0,66
0,0299
0,68
0,0561
APOH_ATVVYQGER_vs_PGRP2_AGLLRPDYALLGHR
48 и 126
0,56
0,4083
0,71
0,0241
B2MG_VEHSDLSFSK_vs_LYAM1_SYYWIGIR
50 и 120
0,7
0,0068
0,61
0,2128
B2MG_VNHVTLSQPK_vs_CRIS3_AVSPPAR
51 и 78
0,74
0,001
0,51
0,8771
B2MG_VNHVTLSQPK_vs_CRIS3_YEDLYSNCK
51 и 79
0,75
7,00E-04
0,51
0,942
B2MG_VNHVTLSQPK_vs_IGF2_GIVEECCFR
51 и 103
0,74
0,0014
0,54
0,6624
B2MG_VNHVTLSQPK_vs_LYAM1_SYYWIGIR
51 и 120
0,72
0,0034
0,66
0,0808
B2MG_VNHVTLSQPK_vs_SHBG_IALGGLLFPASNLR
51 и 18
0,57
0,3602
0,71
0,0241
CATD_VGFAEAAR_vs_ALS_IRPHTFTGLSGLR
57 и 40
0,68
0,015
0,56
0,5423
CATD_VGFAEAAR_vs_C163A_INPASLDK
57 и 54
0,69
0,0114
0,74
0,0103
CATD_VGFAEAAR_vs_CRIS3_AVSPPAR
57 и 78
0,72
0,0038
0,57
0,4449
CATD_VGFAEAAR_vs_CRIS3_YEDLYSNCK
57 и 79
0,73
0,0021
0,57
0,4183
CATD_VGFAEAAR_vs_CSH_AHQLAIDTYQEFEETYIPK
57 и 80
0,67
0,0256
0,69
0,0425
CATD_VGFAEAAR_vs_CSH_ISLLLIESWLEPVR
57 и 81
0,65
0,0392
0,68
0,0505
CATD_VGFAEAAR_vs_FBLN1_TGYYFDGISR
57 и 86
0,65
0,0378
0,65
0,1035
CATD_VGFAEAAR_vs_IBP1_VVESLAK
57 и 97
0,65
0,0428
0,65
0,096
CATD_VGFAEAAR_vs_IBP2_LIQGAPTIR
57 и 98
0,57
0,3353
0,7
0,0318
CATD_VGFAEAAR_vs_IBP3_FLNVLSPR
57 и 99
0,71
0,0057
0,53
0,778
CATD_VGFAEAAR_vs_IBP3_YGQPLPGYTTK
57 и 100
0,71
0,0044
0,54
0,7025
CATD_VGFAEAAR_vs_IGF2_GIVEECCFR
57 и 103
0,75
9,00E-04
0,57
0,4669
CATD_VGFAEAAR_vs_ITIH4_ILDDLSPR
57 и 112
0,67
0,0239
0,58
0,3777
CATD_VGFAEAAR_vs_LYAM1_SYYWIGIR
57 и 120
0,71
0,0054
0,71
0,0247
CATD_VGFAEAAR_vs_PGRP2_AGLLRPDYALLGHR
57 и 126
0,62
0,0991
0,72
0,0148
CATD_VGFAEAAR_vs_PSG3_VSAPSGTGHLPGLNPL
57 и 134
0,66
0,0353
0,65
0,1035
CATD_VGFAEAAR_vs_SHBG_IALGGLLFPASNLR
57 и 18
0,59
0,2231
0,74
0,0098
CATD_VGFAEAAR_vs_SOM2.CSH_NYGLLYCFR
57 и 138
0,66
0,0372
0,67
0,0689
CATD_VGFAEAAR_vs_SOM2.CSH_SVEGSCGF
57 и 139
0,64
0,0634
0,7
0,0293
CATD_VGFAEAAR_vs_SPRL1_VLTHSELAPLR
57 и 140
0,66
0,0292
0,68
0,0454
CATD_VGFAEAAR_vs_TENX_LNWEAPPGAFDSFLLR
57 и 141
0,66
0,0356
0,71
0,0204
CATD_VGFAEAAR_vs_TENX_LSQLSVTDVTTSSLR
57 и 142
0,65
0,0437
0,67
0,0689
CATD_VGFAEAAR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
57 и 144
0,68
0,0163
0,62
0,1965
CATD_VSTLPAITLK_vs_ALS_IRPHTFTGLSGLR
58 и 40
0,68
0,018
0,57
0,4669
CATD_VSTLPAITLK_vs_C163A_INPASLDK
58 и 54
0,66
0,034
0,69
0,039
CATD_VSTLPAITLK_vs_CRIS3_AVSPPAR
58 и 78
0,73
0,0024
0,55
0,5667
CATD_VSTLPAITLK_vs_CRIS3_YEDLYSNCK
58 и 79
0,73
0,0016
0,56
0,4894
CATD_VSTLPAITLK_vs_CSH_AHQLAIDTYQEFEETYIPK
58 и 80
0,66
0,0353
0,65
0,1115
CATD_VSTLPAITLK_vs_FBLN1_TGYYFDGISR
58 и 86
0,66
0,0292
0,63
0,1431
CATD_VSTLPAITLK_vs_IBP1_VVESLAK
58 и 97
0,65
0,0463
0,65
0,0925
CATD_VSTLPAITLK_vs_IBP3_FLNVLSPR
58 и 99
0,71
0,0049
0,55
0,6233
CATD_VSTLPAITLK_vs_IBP3_YGQPLPGYTTK
58 и 100
0,72
0,0032
0,55
0,6105
CATD_VSTLPAITLK_vs_IGF2_GIVEECCFR
58 и 103
0,74
0,0012
0,58
0,3876
CATD_VSTLPAITLK_vs_LYAM1_SYYWIGIR
58 и 120
0,7
0,0071
0,7
0,0277
CATD_VSTLPAITLK_vs_PGRP2_AGLLRPDYALLGHR
58 и 126
0,61
0,1448
0,74
0,0093
CATD_VSTLPAITLK_vs_PSG3_VSAPSGTGHLPGLNPL
58 и 134
0,66
0,0295
0,68
0,055
CATD_VSTLPAITLK_vs_SHBG_IALGGLLFPASNLR
58 и 18
0,59
0,2532
0,75
0,0073
CATD_VSTLPAITLK_vs_SOM2.CSH_NYGLLYCFR
58 и 138
0,65
0,0447
0,62
0,2095
CATD_VSTLPAITLK_vs_SPRL1_VLTHSELAPLR
58 и 140
0,64
0,0557
0,68
0,0484
CATD_VSTLPAITLK_vs_TENX_LNWEAPPGAFDSFLLR
58 и 141
0,65
0,0444
0,73
0,0115
CATD_VSTLPAITLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
58 и 144
0,66
0,0308
0,62
0,1997
CBPN_EALIQFLEQVHQGIK_vs_LYAM1_SYYWIGIR
59 и 120
0,71
0,0055
0,56
0,5244
CBPN_NGVDLNR_vs_LYAM1_SYYWIGIR
157 и 120
0,72
0,0034
0,6
0,2791
CBPN_NGVDLNR_vs_SPRL1_VLTHSELAPLR
157 и 140
0,68
0,0137
0,55
0,5544
CD14_LTVGAAQVPAQLLVGALR_vs_CRIS3_YEDLYSNCK
61 и 79
0,75
9,00E-04
0,52
0,8485
CD14_LTVGAAQVPAQLLVGALR_vs_CSH_AHQLAIDTYQEFEETYIPK
61 и 80
0,68
0,0131
0,54
0,6957
CD14_LTVGAAQVPAQLLVGALR_vs_IBP1_VVESLAK
61 и 97
0,66
0,0301
0,61
0,2371
CD14_LTVGAAQVPAQLLVGALR_vs_IGF2_GIVEECCFR
61 и 103
0,81
0
0,58
0,3876
CD14_LTVGAAQVPAQLLVGALR_vs_LYAM1_SYYWIGIR
61 и 120
0,76
4,00E-04
0,62
0,1934
CD14_LTVGAAQVPAQLLVGALR_vs_PGRP2_AGLLRPDYALLGHR
61 и 126
0,66
0,0364
0,68
0,0484
CD14_LTVGAAQVPAQLLVGALR_vs_PSG3_VSAPSGTGHLPGLNPL
61 и 134
0,67
0,0237
0,56
0,5483
CD14_LTVGAAQVPAQLLVGALR_vs_SHBG_IALGGLLFPASNLR
61 и 18
0,62
0,1048
0,69
0,0349
CD14_LTVGAAQVPAQLLVGALR_vs_SOM2.CSH_SVEGSCGF
61 и 139
0,67
0,0206
0,57
0,4491
CD14_LTVGAAQVPAQLLVGALR_vs_SPRL1_VLTHSELAPLR
61 и 140
0,72
0,0038
0,55
0,5544
CD14_LTVGAAQVPAQLLVGALR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
61 и 144
0,76
6,00E-04
0,52
0,8272
CD14_LTVGAAQVPAQLLVGALR_vs_VTDB_ELPEHTVK
61 и 147
0,73
0,0023
0,52
0,8272
CD14_SWLAELQQWLKPGLK_vs_CRIS3_YEDLYSNCK
62 и 79
0,74
0,0012
0,51
0,9059
CD14_SWLAELQQWLKPGLK_vs_IBP1_VVESLAK
62 и 97
0,66
0,0301
0,6
0,2791
CD14_SWLAELQQWLKPGLK_vs_IGF2_GIVEECCFR
62 и 103
0,79
1,00E-04
0,57
0,4235
CD14_SWLAELQQWLKPGLK_vs_LYAM1_SYYWIGIR
62 и 120
0,75
8,00E-04
0,62
0,2029
CD14_SWLAELQQWLKPGLK_vs_SHBG_IALGGLLFPASNLR
62 и 18
0,61
0,1334
0,68
0,0484
CD14_SWLAELQQWLKPGLK_vs_SOM2.CSH_SVEGSCGF
62 и 139
0,66
0,0297
0,57
0,4714
CD14_SWLAELQQWLKPGLK_vs_SPRL1_VLTHSELAPLR
62 и 140
0,7
0,0069
0,55
0,5979
CD14_SWLAELQQWLKPGLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
62 и 144
0,74
0,0013
0,51
0,942
CD14_SWLAELQQWLKPGLK_vs_VTDB_ELPEHTVK
62 и 147
0,71
0,0047
0,54
0,6691
CFAB_YGLVTYATYPK_vs_LYAM1_SYYWIGIR
64 и 120
0,67
0,0215
0,58
0,3826
CLUS_ASSIIDELFQDR_vs_CRIS3_AVSPPAR
67 и 78
0,71
0,0041
0,53
0,771
CLUS_ASSIIDELFQDR_vs_CRIS3_YEDLYSNCK
67 и 79
0,73
0,0024
0,53
0,716
CLUS_ASSIIDELFQDR_vs_IBP3_YGQPLPGYTTK
67 и 100
0,72
0,0026
0,54
0,6757
CLUS_ASSIIDELFQDR_vs_IGF2_GIVEECCFR
67 и 103
0,74
0,0012
0,52
0,87
CLUS_ASSIIDELFQDR_vs_LYAM1_SYYWIGIR
67 и 120
0,71
0,0056
0,68
0,0484
CLUS_ASSIIDELFQDR_vs_PGRP2_AGLLRPDYALLGHR
67 и 126
0,56
0,4152
0,74
0,0098
CLUS_ASSIIDELFQDR_vs_SHBG_IALGGLLFPASNLR
67 и 18
0,57
0,3539
0,73
0,0115
CLUS_LFDSDPITVTVPVEVSR_vs_CRIS3_AVSPPAR
68 и 78
0,7
0,0059
0,53
0,7296
CLUS_LFDSDPITVTVPVEVSR_vs_CRIS3_YEDLYSNCK
68 и 79
0,72
0,0035
0,55
0,5544
CLUS_LFDSDPITVTVPVEVSR_vs_IBP3_YGQPLPGYTTK
68 и 100
0,72
0,0036
0,53
0,7572
CLUS_LFDSDPITVTVPVEVSR_vs_IGF2_GIVEECCFR
68 и 103
0,73
0,0021
0,52
0,806
CLUS_LFDSDPITVTVPVEVSR_vs_LYAM1_SYYWIGIR
68 и 120
0,67
0,0199
0,68
0,0444
CLUS_LFDSDPITVTVPVEVSR_vs_PGRP2_AGLLRPDYALLGHR
68 и 126
0,55
0,5309
0,73
0,0137
CLUS_LFDSDPITVTVPVEVSR_vs_SHBG_IALGGLLFPASNLR
68 и 18
0,55
0,4899
0,73
0,0106
CO5_TLLPVSKPEIR_vs_CRIS3_YEDLYSNCK
70 и 79
0,73
0,0023
0,5
1
CO5_TLLPVSKPEIR_vs_LYAM1_SYYWIGIR
70 и 120
0,69
0,0089
0,68
0,0538
CO5_TLLPVSKPEIR_vs_SHBG_IALGGLLFPASNLR
70 и 18
0,54
0,5469
0,72
0,0164
CO5_VFQFLEK_vs_LYAM1_SYYWIGIR
71 и 120
0,69
0,0108
0,62
0,1873
CO6_ALNHLPLEYNSALYSR_vs_LYAM1_SYYWIGIR
72 и 120
0,7
0,0069
0,65
0,1074
CO6_ALNHLPLEYNSALYSR_vs_SHBG_IALGGLLFPASNLR
72 и 18
0,55
0,5053
0,72
0,0176
CO8A_SLLQPNK_vs_IGF2_GIVEECCFR
74 и 103
0,73
0,002
0,55
0,6233
CO8A_SLLQPNK_vs_LYAM1_SYYWIGIR
74 и 120
0,71
0,0049
0,62
0,1842
CO8B_QALEEFQK_vs_IGF2_GIVEECCFR
76 и 103
0,72
0,0037
0,52
0,792
CO8B_QALEEFQK_vs_LYAM1_SYYWIGIR
76 и 120
0,67
0,0194
0,62
0,1965
F13B_GDTYPAELYITGSILR_vs_CRIS3_AVSPPAR
84 и 78
0,73
0,0018
0,5
0,9927
F13B_GDTYPAELYITGSILR_vs_CRIS3_YEDLYSNCK
84 и 79
0,74
0,0012
0,52
0,792
F13B_GDTYPAELYITGSILR_vs_IBP3_YGQPLPGYTTK
84 и 100
0,74
0,0015
0,56
0,4952
F13B_GDTYPAELYITGSILR_vs_IGF2_GIVEECCFR
84 и 103
0,75
7,00E-04
0,51
0,8771
F13B_GDTYPAELYITGSILR_vs_LYAM1_SYYWIGIR
84 и 120
0,72
0,0026
0,68
0,0538
F13B_GDTYPAELYITGSILR_vs_PGRP2_AGLLRPDYALLGHR
84 и 126
0,59
0,2445
0,69
0,0407
F13B_GDTYPAELYITGSILR_vs_SHBG_IALGGLLFPASNLR
84 и 18
0,57
0,3445
0,7
0,0265
F13B_GDTYPAELYITGSILR_vs_SPRL1_VLTHSELAPLR
84 и 140
0,65
0,0392
0,61
0,2335
FETUA_FSVVYAK_vs_CRIS3_YEDLYSNCK
88 и 79
0,7
0,0069
0,53
0,7572
FETUA_FSVVYAK_vs_LYAM1_SYYWIGIR
88 и 120
0,67
0,0258
0,67
0,0689
FETUA_HTLNQIDEVK_vs_LYAM1_SYYWIGIR
89 и 120
0,67
0,022
0,61
0,2128
HEMO_NFPSPVDAAFR_vs_LYAM1_SYYWIGIR
93 и 120
0,66
0,0271
0,64
0,1335
IBP4_QCHPALDGQR_vs_CRIS3_AVSPPAR
2 и 78
0,75
8,00E-04
0,51
0,9348
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,76
4,00E-04
0,51
0,8987
IBP4_QCHPALDGQR_vs_IGF2_GIVEECCFR
2 и 103
0,71
0,0039
0,52
0,806
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,7
0,0078
0,67
0,0649
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,56
0,4544
0,71
0,023
IBP6_HLDSVLQQLQTEVYR_vs_CRIS3_YEDLYSNCK
102 и 79
0,75
6,00E-04
0,57
0,4235
IBP6_HLDSVLQQLQTEVYR_vs_LYAM1_SYYWIGIR
102 и 120
0,7
0,0067
0,54
0,6559
KNG1_DIPTNSPELEETLTHTITK_vs_LYAM1_SYYWIGIR
116 и 120
0,7
0,008
0,65
0,1135
KNG1_QVVAGLNFR_vs_CRIS3_AVSPPAR
117 и 78
0,72
0,0035
0,51
0,8987
KNG1_QVVAGLNFR_vs_CRIS3_YEDLYSNCK
117 и 79
0,73
0,0023
0,54
0,6691
KNG1_QVVAGLNFR_vs_IGF2_GIVEECCFR
117 и 103
0,76
4,00E-04
0,53
0,7572
KNG1_QVVAGLNFR_vs_ITIH4_ILDDLSPR
117 и 112
0,69
0,0121
0,54
0,6559
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,73
0,0021
0,65
0,0925
KNG1_QVVAGLNFR_vs_PGRP2_AGLLRPDYALLGHR
117 и 126
0,61
0,1533
0,7
0,0333
KNG1_QVVAGLNFR_vs_SHBG_IALGGLLFPASNLR
117 и 18
0,59
0,2433
0,7
0,0259
LBP_ITLPDFTGDLR_vs_LYAM1_SYYWIGIR
119 и 120
0,71
0,0055
0,55
0,5728
PEDF_LQSLFDSPDFSK_vs_CRIS3_AVSPPAR
124 и 78
0,74
0,0011
0,52
0,799
PEDF_LQSLFDSPDFSK_vs_CRIS3_YEDLYSNCK
124 и 79
0,75
8,00E-04
0,52
0,8557
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,7
0,0062
0,63
0,156
PEDF_TVQAVLTVPK_vs_CRIS3_AVSPPAR
125 и 78
0,75
9,00E-04
0,52
0,8343
PEDF_TVQAVLTVPK_vs_CRIS3_YEDLYSNCK
125 и 79
0,76
6,00E-04
0,5
0,9927
PEDF_TVQAVLTVPK_vs_IGF2_GIVEECCFR
125 и 103
0,77
3,00E-04
0,58
0,3876
PEDF_TVQAVLTVPK_vs_LYAM1_SYYWIGIR
125 и 120
0,72
0,0033
0,64
0,1156
PSG2_IHPSYTNYR_vs_CRIS3_AVSPPAR
133 и 78
0,72
0,0036
0,54
0,6757
PSG2_IHPSYTNYR_vs_CRIS3_YEDLYSNCK
133 и 79
0,72
0,0033
0,55
0,5728
PSG2_IHPSYTNYR_vs_FBLN1_TGYYFDGISR
133 и 86
0,67
0,0189
0,59
0,3215
PSG2_IHPSYTNYR_vs_IBP1_VVESLAK
133 и 97
0,66
0,0372
0,58
0,3583
PSG2_IHPSYTNYR_vs_IGF2_GIVEECCFR
133 и 103
0,69
0,0123
0,52
0,8414
PSG2_IHPSYTNYR_vs_LYAM1_SYYWIGIR
133 и 120
0,7
0,008
0,62
0,1965
PTGDS_GPGEDFR_vs_CRIS3_YEDLYSNCK
137 и 79
0,73
0,0023
0,52
0,8343
PTGDS_GPGEDFR_vs_LYAM1_SYYWIGIR
137 и 120
0,67
0,0224
0,61
0,2299
THBG_AVLHIGEK_vs_LYAM1_SYYWIGIR
143 и 120
0,69
0,0106
0,59
0,3215
VTNC_GQYCYELDEK_vs_CRIS3_YEDLYSNCK
149 и 79
0,7
0,0066
0,54
0,6298
VTNC_GQYCYELDEK_vs_IGF2_GIVEECCFR
149 и 103
0,7
0,0064
0,54
0,6823
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,68
0,0136
0,7
0,0297
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,54
0,6305
0,75
0,0065
VTNC_GQYCYELDEK_vs_PSG3_VSAPSGTGHLPGLNPL
149 и 134
0,59
0,2185
0,7
0,0311
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,55
0,4842
0,73
0,0137
VTNC_VDTVDPPYPR_vs_CRIS3_YEDLYSNCK
150 и 79
0,69
0,0095
0,55
0,6169
VTNC_VDTVDPPYPR_vs_IGF2_GIVEECCFR
150 и 103
0,68
0,0139
0,53
0,7434
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,66
0,0361
0,72
0,0168
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,54
0,5818
0,73
0,0121
Таблица 68: эффективность классификации по обращениям, недели 17-21.
AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, с BMI-стратификацией (>22≤37), отдельно для первой беременности и повторной беременности.
Обращение
SEQ ID №
119_153_rBMI_35 multi ROC_AUC
119_153_rBMI_35 multi P-значение
119_153_rBMI_35 primi ROC_AUC
119_153_rBMI_35 primi P-значение
A2GL_DLLLPQPDLR_vs_CRIS3_YEDLYSNCK
34 и 79
0,79
0,0013
0,51
0,9432
A2GL_DLLLPQPDLR_vs_LYAM1_SYYWIGIR
34 и 120
0,73
0,0091
0,68
0,1013
AFAM_DADPDTFFAK_vs_CRIS3_YEDLYSNCK
37 и 79
0,74
0,0073
0,56
0,5959
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,79
0,0015
0,51
0,9432
AFAM_DADPDTFFAK_vs_LYAM1_SYYWIGIR
37 и 120
0,69
0,0371
0,71
0,0525
AFAM_DADPDTFFAK_vs_PGRP2_AGLLRPDYALLGHR
37 и 126
0,63
0,1361
0,72
0,047
AFAM_HFQNLGK_vs_ALS_IRPHTFTGLSGLR
38 и 40
0,75
0,0048
0,54
0,7456
AFAM_HFQNLGK_vs_CRIS3_AVSPPAR
38 и 78
0,72
0,015
0,56
0,585
AFAM_HFQNLGK_vs_CRIS3_YEDLYSNCK
38 и 79
0,75
0,0062
0,57
0,5215
AFAM_HFQNLGK_vs_IBP3_FLNVLSPR
38 и 99
0,75
0,0057
0,51
0,9558
AFAM_HFQNLGK_vs_IBP3_YGQPLPGYTTK
38 и 100
0,78
0,0022
0,54
0,7099
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,81
7,00E-04
0,5
0,9811
AFAM_HFQNLGK_vs_LYAM1_SYYWIGIR
38 и 120
0,68
0,0404
0,72
0,0388
AFAM_HFQNLGK_vs_PGRP2_AGLLRPDYALLGHR
38 и 126
0,64
0,115
0,72
0,0436
ANGT_DPTFIPAPIQAK_vs_CRIS3_AVSPPAR
42 и 78
0,79
0,0012
0,51
0,9684
ANGT_DPTFIPAPIQAK_vs_CRIS3_YEDLYSNCK
42 и 79
0,8
9,00E-04
0,52
0,893
ANGT_DPTFIPAPIQAK_vs_LYAM1_SYYWIGIR
42 и 120
0,76
0,0037
0,67
0,1152
ANGT_DPTFIPAPIQAK_vs_SOM2.CSH_SVEGSCGF
42 и 139
0,7
0,0293
0,57
0,5528
APOH_ATVVYQGER_vs_CRIS3_YEDLYSNCK
48 и 79
0,73
0,0107
0,59
0,4241
APOH_ATVVYQGER_vs_LYAM1_SYYWIGIR
48 и 120
0,65
0,0888
0,73
0,032
APOH_ATVVYQGER_vs_PGRP2_AGLLRPDYALLGHR
48 и 126
0,6
0,283
0,77
0,0116
B2MG_VNHVTLSQPK_vs_CRIS3_AVSPPAR
51 и 78
0,76
0,0038
0,54
0,7099
B2MG_VNHVTLSQPK_vs_CRIS3_YEDLYSNCK
51 и 79
0,77
0,0028
0,56
0,585
B2MG_VNHVTLSQPK_vs_LYAM1_SYYWIGIR
51 и 120
0,71
0,0201
0,75
0,0213
CATD_VGFAEAAR_vs_C163A_INPASLDK
57 и 54
0,69
0,0387
0,8
0,0057
CATD_VGFAEAAR_vs_CRIS3_AVSPPAR
57 и 78
0,7
0,0256
0,66
0,1431
CATD_VGFAEAAR_vs_CRIS3_YEDLYSNCK
57 и 79
0,72
0,0126
0,66
0,152
CATD_VGFAEAAR_vs_CSH_AHQLAIDTYQEFEETYIPK
57 и 80
0,7
0,0292
0,7
0,0629
CATD_VGFAEAAR_vs_CSH_ISLLLIESWLEPVR
57 и 81
0,68
0,0409
0,7
0,0629
CATD_VGFAEAAR_vs_IBP3_FLNVLSPR
57 и 99
0,69
0,0346
0,63
0,2259
CATD_VGFAEAAR_vs_IBP3_YGQPLPGYTTK
57 и 100
0,68
0,0404
0,62
0,2782
CATD_VGFAEAAR_vs_IGF2_GIVEECCFR
57 и 103
0,74
0,0077
0,62
0,2853
CATD_VGFAEAAR_vs_LYAM1_SYYWIGIR
57 и 120
0,69
0,0341
0,82
0,0037
CATD_VGFAEAAR_vs_PGRP2_AGLLRPDYALLGHR
57 и 126
0,61
0,2372
0,83
0,0027
CATD_VGFAEAAR_vs_SHBG_IALGGLLFPASNLR
57 и 18
0,58
0,3917
0,78
0,0092
CATD_VGFAEAAR_vs_SOM2.CSH_NYGLLYCFR
57 и 138
0,68
0,0409
0,66
0,1388
CATD_VGFAEAAR_vs_SPRL1_VLTHSELAPLR
57 и 140
0,62
0,167
0,73
0,0374
CATD_VGFAEAAR_vs_TENX_LNWEAPPGAFDSFLLR
57 и 141
0,62
0,1968
0,73
0,0307
CATD_VSTLPAITLK_vs_C163A_INPASLDK
58 и 54
0,66
0,0685
0,76
0,0165
CATD_VSTLPAITLK_vs_CRIS3_AVSPPAR
58 и 78
0,73
0,01
0,66
0,1475
CATD_VSTLPAITLK_vs_CRIS3_YEDLYSNCK
58 и 79
0,75
0,0057
0,67
0,1189
CATD_VSTLPAITLK_vs_CSH_AHQLAIDTYQEFEETYIPK
58 и 80
0,7
0,0275
0,67
0,1152
CATD_VSTLPAITLK_vs_CSH_ISLLLIESWLEPVR
58 и 81
0,68
0,0426
0,67
0,1266
CATD_VSTLPAITLK_vs_IBP3_FLNVLSPR
58 и 99
0,71
0,0217
0,68
0,0949
CATD_VSTLPAITLK_vs_IBP3_YGQPLPGYTTK
58 и 100
0,7
0,0287
0,65
0,1565
CATD_VSTLPAITLK_vs_IGF2_GIVEECCFR
58 и 103
0,73
0,0098
0,66
0,1306
CATD_VSTLPAITLK_vs_LYAM1_SYYWIGIR
58 и 120
0,69
0,0304
0,83
0,0026
CATD_VSTLPAITLK_vs_PGRP2_AGLLRPDYALLGHR
58 и 126
0,61
0,226
0,85
0,0012
CATD_VSTLPAITLK_vs_PSG3_VSAPSGTGHLPGLNPL
58 и 134
0,64
0,1317
0,72
0,0404
CATD_VSTLPAITLK_vs_SHBG_IALGGLLFPASNLR
58 и 18
0,58
0,3639
0,81
0,0043
CATD_VSTLPAITLK_vs_SOM2.CSH_NYGLLYCFR
58 и 138
0,69
0,0327
0,62
0,2853
CATD_VSTLPAITLK_vs_TENX_LNWEAPPGAFDSFLLR
58 и 141
0,63
0,1584
0,77
0,0121
CBPN_EALIQFLEQVHQGIK_vs_LYAM1_SYYWIGIR
59 и 120
0,71
0,0186
0,63
0,2199
CBPN_NGVDLNR_vs_CRIS3_YEDLYSNCK
157 и 79
0,77
0,0023
0,52
0,8805
CBPN_NGVDLNR_vs_LYAM1_SYYWIGIR
157 и 120
0,73
0,0094
0,68
0,1047
CD14_LTVGAAQVPAQLLVGALR_vs_CRIS3_AVSPPAR
61 и 78
0,77
0,0029
0,52
0,893
CD14_LTVGAAQVPAQLLVGALR_vs_CRIS3_YEDLYSNCK
61 и 79
0,78
0,0022
0,52
0,8308
CD14_LTVGAAQVPAQLLVGALR_vs_LYAM1_SYYWIGIR
61 и 120
0,76
0,0043
0,71
0,0585
CD14_LTVGAAQVPAQLLVGALR_vs_PGRP2_AGLLRPDYALLGHR
61 и 126
0,68
0,0444
0,72
0,0452
CD14_LTVGAAQVPAQLLVGALR_vs_SOM2.CSH_SVEGSCGF
61 и 139
0,75
0,0057
0,51
0,9055
CD14_SWLAELQQWLKPGLK_vs_LYAM1_SYYWIGIR
62 и 120
0,74
0,0082
0,67
0,1116
CFAB_YGLVTYATYPK_vs_LYAM1_SYYWIGIR
64 и 120
0,68
0,0463
0,64
0,2026
CLUS_ASSIIDELFQDR_vs_CRIS3_AVSPPAR
67 и 78
0,75
0,0051
0,54
0,6865
CLUS_ASSIIDELFQDR_vs_CRIS3_YEDLYSNCK
67 и 79
0,76
0,0035
0,57
0,5528
CLUS_ASSIIDELFQDR_vs_LYAM1_SYYWIGIR
67 и 120
0,7
0,0292
0,74
0,0295
CLUS_ASSIIDELFQDR_vs_PGRP2_AGLLRPDYALLGHR
67 и 126
0,59
0,3461
0,74
0,0295
CLUS_LFDSDPITVTVPVEVSR_vs_CRIS3_YEDLYSNCK
68 и 79
0,74
0,0088
0,57
0,5215
CLUS_LFDSDPITVTVPVEVSR_vs_LYAM1_SYYWIGIR
68 и 120
0,62
0,1871
0,73
0,0374
CO5_TLLPVSKPEIR_vs_CRIS3_YEDLYSNCK
70 и 79
0,76
0,0042
0,52
0,8805
CO5_TLLPVSKPEIR_vs_LYAM1_SYYWIGIR
70 и 120
0,7
0,0252
0,74
0,0283
CO6_ALNHLPLEYNSALYSR_vs_LYAM1_SYYWIGIR
72 и 120
0,71
0,0207
0,73
0,0307
CO6_ALNHLPLEYNSALYSR_vs_PGRP2_AGLLRPDYALLGHR
72 и 126
0,61
0,226
0,76
0,0172
CO8A_SLLQPNK_vs_LYAM1_SYYWIGIR
74 и 120
0,71
0,0173
0,67
0,1116
F13B_GDTYPAELYITGSILR_vs_CRIS3_AVSPPAR
84 и 78
0,78
0,0021
0,54
0,6981
F13B_GDTYPAELYITGSILR_vs_CRIS3_YEDLYSNCK
84 и 79
0,79
0,0015
0,56
0,5741
F13B_GDTYPAELYITGSILR_vs_IGF2_GIVEECCFR
84 и 103
0,78
0,0017
0,55
0,6405
F13B_GDTYPAELYITGSILR_vs_LYAM1_SYYWIGIR
84 и 120
0,74
0,0081
0,74
0,0295
F13B_GDTYPAELYITGSILR_vs_PGRP2_AGLLRPDYALLGHR
84 и 126
0,63
0,1346
0,72
0,0404
FETUA_FSVVYAK_vs_LYAM1_SYYWIGIR
88 и 120
0,65
0,0989
0,74
0,0283
FETUA_HTLNQIDEVK_vs_LYAM1_SYYWIGIR
89 и 120
0,66
0,0703
0,72
0,0436
HEMO_NFPSPVDAAFR_vs_LYAM1_SYYWIGIR
93 и 120
0,62
0,1705
0,74
0,0241
IBP4_QCHPALDGQR_vs_CRIS3_AVSPPAR
2 и 78
0,74
0,0075
0,57
0,5011
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,77
0,0027
0,59
0,4241
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,7
0,0283
0,78
0,0111
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,56
0,5187
0,73
0,036
IBP6_HLDSVLQQLQTEVYR_vs_LYAM1_SYYWIGIR
102 и 120
0,73
0,0098
0,6
0,3711
ITIH3_ALDLSLK_vs_LYAM1_SYYWIGIR
111 и 120
0,76
0,0045
0,56
0,585
ITIH3_ALDLSLK_vs_SHBG_IALGGLLFPASNLR
111 и 18
0,68
0,0475
0,58
0,4911
KNG1_DIPTNSPELEETLTHTITK_vs_LYAM1_SYYWIGIR
116 и 120
0,67
0,0557
0,74
0,0251
KNG1_QVVAGLNFR_vs_CRIS3_AVSPPAR
117 и 78
0,73
0,011
0,59
0,4241
KNG1_QVVAGLNFR_vs_CRIS3_YEDLYSNCK
117 и 79
0,74
0,0079
0,61
0,3148
KNG1_QVVAGLNFR_vs_IGF2_GIVEECCFR
117 и 103
0,78
0,0016
0,52
0,8431
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,71
0,022
0,76
0,0158
KNG1_QVVAGLNFR_vs_PGRP2_AGLLRPDYALLGHR
117 и 126
0,6
0,2655
0,75
0,0204
PAPP1_DIPHWLNPTR_vs_CRIS3_AVSPPAR
122 и 78
0,72
0,0148
0,58
0,4812
PAPP1_DIPHWLNPTR_vs_CRIS3_YEDLYSNCK
122 и 79
0,72
0,0134
0,59
0,3883
PAPP1_DIPHWLNPTR_vs_SOM2.CSH_SVEGSCGF
122 и 139
0,68
0,043
0,68
0,1047
PEDF_LQSLFDSPDFSK_vs_CRIS3_YEDLYSNCK
124 и 79
0,78
0,0021
0,51
0,9684
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,75
0,0062
0,66
0,152
PEDF_TVQAVLTVPK_vs_CRIS3_AVSPPAR
125 и 78
0,78
0,0022
0,5
0,9811
PEDF_TVQAVLTVPK_vs_CRIS3_YEDLYSNCK
125 и 79
0,79
0,0015
0,52
0,8555
PEDF_TVQAVLTVPK_vs_LYAM1_SYYWIGIR
125 и 120
0,75
0,0065
0,68
0,0949
PSG2_IHPSYTNYR_vs_ALS_IRPHTFTGLSGLR
133 и 40
0,68
0,0482
0,62
0,2853
PSG2_IHPSYTNYR_vs_CRIS3_AVSPPAR
133 и 78
0,78
0,0022
0,6
0,3463
PSG2_IHPSYTNYR_vs_CRIS3_YEDLYSNCK
133 и 79
0,78
0,0022
0,61
0,2998
PSG2_IHPSYTNYR_vs_CSH_AHQLAIDTYQEFEETYIPK
133 и 80
0,69
0,0393
0,6
0,3382
PSG2_IHPSYTNYR_vs_CSH_ISLLLIESWLEPVR
133 и 81
0,68
0,0438
0,58
0,4714
PSG2_IHPSYTNYR_vs_IBP3_FLNVLSPR
133 и 99
0,7
0,023
0,58
0,4426
PSG2_IHPSYTNYR_vs_IBP3_YGQPLPGYTTK
133 и 100
0,7
0,0237
0,58
0,4812
PSG2_IHPSYTNYR_vs_IGF2_GIVEECCFR
133 и 103
0,73
0,0105
0,57
0,5422
PSG2_IHPSYTNYR_vs_ITIH4_ILDDLSPR
133 и 112
0,69
0,0361
0,64
0,197
PSG2_IHPSYTNYR_vs_LYAM1_SYYWIGIR
133 и 120
0,75
0,0054
0,72
0,0452
PSG2_IHPSYTNYR_vs_PGRP2_AGLLRPDYALLGHR
133 и 126
0,69
0,0356
0,69
0,0776
PSG2_IHPSYTNYR_vs_SHBG_IALGGLLFPASNLR
133 и 18
0,69
0,0361
0,72
0,0452
PSG2_IHPSYTNYR_vs_SOM2.CSH_SVEGSCGF
133 и 139
0,71
0,0202
0,62
0,2853
PTGDS_GPGEDFR_vs_CRIS3_AVSPPAR
137 и 78
0,75
0,0057
0,53
0,8062
PTGDS_GPGEDFR_vs_CRIS3_YEDLYSNCK
137 и 79
0,76
0,0038
0,55
0,6519
PTGDS_GPGEDFR_vs_LYAM1_SYYWIGIR
137 и 120
0,68
0,0409
0,7
0,0724
VTNC_GQYCYELDEK_vs_CRIS3_AVSPPAR
149 и 78
0,72
0,0157
0,59
0,3971
VTNC_GQYCYELDEK_vs_CRIS3_YEDLYSNCK
149 и 79
0,74
0,0078
0,6
0,3544
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,7
0,0292
0,8
0,0052
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,56
0,486
0,79
0,0066
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,56
0,4824
0,74
0,0272
VTNC_VDTVDPPYPR_vs_CRIS3_YEDLYSNCK
150 и 79
0,7
0,0241
0,61
0,3225
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,63
0,1518
0,82
0,0032
Таблица 69: эффективность классификации по обращениям, недели 17-21.
AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, без BMI-стратификации, отдельно по полу плода.
Обращение
SEQ ID №
119_153_aBMI_37 жен. ROC_AUC
119_153_aBMI_37 жен. P-значение
119_153_aBMI_37 муж. ROC_AUC
119_153_aBMI_37 муж. P-значение
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,56
0,2281
0,66
0,001
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,58
0,1136
0,65
0,0019
AFAM_HFQNLGK_vs_PSG3_VSAPSGTGHLPGLNPL
38 и 134
0,54
0,5033
0,67
3,00E-04
APOC3_GWVTDGFSSLK_vs_IBP3_FLNVLSPR
47 и 99
0,61
0,0344
0,65
0,0024
APOC3_GWVTDGFSSLK_vs_IGF2_GIVEECCFR
47 и 103
0,61
0,0397
0,66
9,00E-04
APOC3_GWVTDGFSSLK_vs_PSG3_VSAPSGTGHLPGLNPL
47 и 134
0,62
0,0235
0,66
7,00E-04
APOC3_GWVTDGFSSLK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
47 и 135
0,57
0,1935
0,65
0,0025
APOH_ATVVYQGER_vs_IGF2_GIVEECCFR
48 и 103
0,54
0,4302
0,67
4,00E-04
APOH_ATVVYQGER_vs_PSG3_VSAPSGTGHLPGLNPL
48 и 134
0,53
0,6338
0,69
1,00E-04
APOH_ATVVYQGER_vs_SHBG_IALGGLLFPASNLR
48 и 18
0,53
0,5575
0,67
5,00E-04
C1QB_VPGLYYFTYHASSR_vs_TENX_LNWEAPPGAFDSFLLR
55 и 141
0,66
0,003
0,56
0,1846
CATD_VGFAEAAR_vs_PSG3_VSAPSGTGHLPGLNPL
57 и 134
0,56
0,2984
0,66
0,0012
CATD_VGFAEAAR_vs_SHBG_IALGGLLFPASNLR
57 и 18
0,54
0,4632
0,65
0,0022
CD14_LTVGAAQVPAQLLVGALR_vs_TENX_LNWEAPPGAFDSFLLR
61 и 141
0,66
0,0035
0,56
0,199
CD14_SWLAELQQWLKPGLK_vs_TENX_LNWEAPPGAFDSFLLR
62 и 141
0,66
0,0025
0,56
0,2459
CFAB_YGLVTYATYPK_vs_PGRP2_AGLLRPDYALLGHR
64 и 126
0,65
0,0043
0,57
0,1591
CFAB_YGLVTYATYPK_vs_PSG3_VSAPSGTGHLPGLNPL
64 и 134
0,61
0,0329
0,66
9,00E-04
CFAB_YGLVTYATYPK_vs_TENX_LNWEAPPGAFDSFLLR
64 и 141
0,67
0,0015
0,57
0,1549
CO5_TLLPVSKPEIR_vs_TENX_LNWEAPPGAFDSFLLR
70 и 141
0,67
0,0016
0,55
0,334
CO5_VFQFLEK_vs_TENX_LNWEAPPGAFDSFLLR
71 и 141
0,66
0,0037
0,55
0,2611
CO8A_SLLQPNK_vs_TENX_LNWEAPPGAFDSFLLR
74 и 141
0,66
0,0025
0,58
0,0994
CO8B_QALEEFQK_vs_IGF2_GIVEECCFR
76 и 103
0,57
0,1956
0,66
8,00E-04
CO8B_QALEEFQK_vs_PSG3_VSAPSGTGHLPGLNPL
76 и 134
0,59
0,0972
0,65
0,002
FETUA_FSVVYAK_vs_TENX_LNWEAPPGAFDSFLLR
88 и 141
0,67
0,0019
0,56
0,1782
FETUA_HTLNQIDEVK_vs_PSG3_VSAPSGTGHLPGLNPL
89 и 134
0,57
0,1999
0,65
0,0014
FETUA_HTLNQIDEVK_vs_TENX_LNWEAPPGAFDSFLLR
89 и 141
0,65
0,0044
0,58
0,096
HABP2_FLNWIK_vs_TENX_LNWEAPPGAFDSFLLR
92 и 141
0,67
0,0016
0,58
0,1096
HABP2_FLNWIK_vs_TENX_LSQLSVTDVTTSSLR
92 и 142
0,66
0,0033
0,55
0,254
HEMO_NFPSPVDAAFR_vs_TENX_LNWEAPPGAFDSFLLR
93 и 141
0,66
0,0034
0,56
0,2346
IBP4_QCHPALDGQR_vs_IGF2_GIVEECCFR
2 и 103
0,59
0,0866
0,65
0,0018
IBP4_QCHPALDGQR_vs_PSG3_VSAPSGTGHLPGLNPL
2 и 134
0,58
0,1291
0,7
0
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,58
0,1611
0,68
1,00E-04
IBP4_QCHPALDGQR_vs_TENX_LNWEAPPGAFDSFLLR
2 и 141
0,65
0,0064
0,59
0,049
INHBC_LDFHFSSDR_vs_IBP3_FLNVLSPR
107 и 99
0,55
0,307
0,67
4,00E-04
INHBC_LDFHFSSDR_vs_IBP3_YGQPLPGYTTK
107 и 100
0,57
0,2257
0,67
5,00E-04
INHBC_LDFHFSSDR_vs_IGF2_GIVEECCFR
107 и 103
0,57
0,1988
0,7
0
INHBC_LDFHFSSDR_vs_ITIH4_ILDDLSPR
107 и 112
0,53
0,5951
0,66
9,00E-04
INHBC_LDFHFSSDR_vs_PSG3_VSAPSGTGHLPGLNPL
107 и 134
0,56
0,2695
0,7
0
INHBC_LDFHFSSDR_vs_SHBG_IALGGLLFPASNLR
107 и 18
0,54
0,4196
0,68
2,00E-04
INHBC_LDFHFSSDR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
107 и 144
0,56
0,2577
0,65
0,0018
INHBC_LDFHFSSDR_vs_VTDB_ELPEHTVK
107 и 147
0,54
0,4632
0,68
2,00E-04
ITIH3_ALDLSLK_vs_SHBG_IALGGLLFPASNLR
111 и 18
0,59
0,0821
0,65
0,0017
KNG1_DIPTNSPELEETLTHTITK_vs_PSG3_VSAPSGTGHLPGLNPL
116 и 134
0,56
0,2885
0,66
0,001
LBP_ITGFLKPGK_vs_CHL1_VIAVNEVGR
118 и 66
0,65
0,0056
0,56
0,1773
LBP_ITGFLKPGK_vs_IGF2_GIVEECCFR
118 и 103
0,66
0,0037
0,58
0,0905
LBP_ITGFLKPGK_vs_PGRP2_AGLLRPDYALLGHR
118 и 126
0,65
0,004
0,56
0,2517
LBP_ITGFLKPGK_vs_PSG3_VSAPSGTGHLPGLNPL
118 и 134
0,65
0,0046
0,63
0,0063
LBP_ITGFLKPGK_vs_TENX_LNWEAPPGAFDSFLLR
118 и 141
0,66
0,0032
0,55
0,2563
LBP_ITLPDFTGDLR_vs_CHL1_VIAVNEVGR
119 и 66
0,67
0,0019
0,58
0,0905
LBP_ITLPDFTGDLR_vs_CRIS3_YEDLYSNCK
119 и 79
0,65
0,0049
0,6
0,0403
LBP_ITLPDFTGDLR_vs_IBP3_FLNVLSPR
119 и 99
0,66
0,0037
0,57
0,1693
LBP_ITLPDFTGDLR_vs_IBP3_YGQPLPGYTTK
119 и 100
0,67
0,0017
0,57
0,1667
LBP_ITLPDFTGDLR_vs_IGF2_GIVEECCFR
119 и 103
0,68
0,0011
0,59
0,0503
LBP_ITLPDFTGDLR_vs_LYAM1_SYYWIGIR
119 и 120
0,66
0,0024
0,6
0,0414
LBP_ITLPDFTGDLR_vs_PGRP2_AGLLRPDYALLGHR
119 и 126
0,68
6,00E-04
0,57
0,1746
LBP_ITLPDFTGDLR_vs_PSG3_VSAPSGTGHLPGLNPL
119 и 134
0,66
0,0025
0,65
0,0021
LBP_ITLPDFTGDLR_vs_TENX_LNWEAPPGAFDSFLLR
119 и 141
0,68
0,001
0,57
0,1241
LBP_ITLPDFTGDLR_vs_TENX_LSQLSVTDVTTSSLR
119 и 142
0,67
0,0013
0,55
0,2757
LBP_ITLPDFTGDLR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
119 и 144
0,66
0,0032
0,57
0,1276
LBP_ITLPDFTGDLR_vs_VTDB_ELPEHTVK
119 и 147
0,67
0,0015
0,59
0,0516
PEDF_LQSLFDSPDFSK_vs_PSG3_VSAPSGTGHLPGLNPL
124 и 134
0,57
0,1704
0,67
3,00E-04
PEDF_LQSLFDSPDFSK_vs_TENX_LNWEAPPGAFDSFLLR
124 и 141
0,65
0,0056
0,59
0,058
PEDF_TVQAVLTVPK_vs_PSG3_VSAPSGTGHLPGLNPL
125 и 134
0,57
0,2234
0,66
7,00E-04
VTNC_GQYCYELDEK_vs_ALS_IRPHTFTGLSGLR
149 и 40
0,55
0,3415
0,65
0,0022
VTNC_GQYCYELDEK_vs_IGF2_GIVEECCFR
149 и 103
0,6
0,0581
0,67
5,00E-04
VTNC_GQYCYELDEK_vs_PSG3_VSAPSGTGHLPGLNPL
149 и 134
0,59
0,0871
0,69
1,00E-04
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,59
0,0774
0,68
2,00E-04
VTNC_GQYCYELDEK_vs_VTDB_ELPEHTVK
149 и 147
0,59
0,0805
0,66
0,001
VTNC_VDTVDPPYPR_vs_IGF2_GIVEECCFR
150 и 103
0,59
0,0871
0,66
7,00E-04
VTNC_VDTVDPPYPR_vs_NCAM1_GLGEISAASEFK
150 и 121
0,54
0,4502
0,65
0,0017
VTNC_VDTVDPPYPR_vs_PSG3_VSAPSGTGHLPGLNPL
150 и 134
0,58
0,137
0,7
0
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,59
0,1055
0,67
3,00E-04
VTNC_VDTVDPPYPR_vs_VTDB_ELPEHTVK
150 и 147
0,6
0,0523
0,68
2,00E-04
Таблица 70: эффективность классификации по обращениям, недели 17-21.
AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, с BMI-стратификацией (>22≤37), отдельно по полу плода.
Обращение
SEQ ID №
119_153_rBMI_37 жен. ROC_AUC
119_153_rBMI_37 жен. P-значение
119_153_rBMI_37 муж. ROC_AUC
119_153_rBMI_37 муж. P-значение
A2GL_DLLLPQPDLR_vs_IGF2_GIVEECCFR
34 и 103
0,65
0,0229
0,6
0,0902
A2GL_DLLLPQPDLR_vs_PGRP2_AGLLRPDYALLGHR
34 и 126
0,66
0,0133
0,56
0,3335
AFAM_DADPDTFFAK_vs_IGF2_GIVEECCFR
37 и 103
0,62
0,069
0,65
0,0081
AFAM_DADPDTFFAK_vs_PSG3_VSAPSGTGHLPGLNPL
37 и 134
0,52
0,7127
0,66
0,0057
AFAM_HFQNLGK_vs_IBP3_YGQPLPGYTTK
38 и 100
0,66
0,0169
0,63
0,026
AFAM_HFQNLGK_vs_PSG3_VSAPSGTGHLPGLNPL
38 и 134
0,55
0,4359
0,66
0,0069
ANGT_DPTFIPAPIQAK_vs_CHL1_VIAVNEVGR
42 и 66
0,67
0,0087
0,55
0,382
ANGT_DPTFIPAPIQAK_vs_CRIS3_AVSPPAR
42 и 78
0,68
0,0059
0,57
0,1945
ANGT_DPTFIPAPIQAK_vs_CRIS3_YEDLYSNCK
42 и 79
0,68
0,0048
0,58
0,1677
ANGT_DPTFIPAPIQAK_vs_IGF2_GIVEECCFR
42 и 103
0,65
0,0197
0,59
0,1059
ANGT_DPTFIPAPIQAK_vs_LYAM1_SYYWIGIR
42 и 120
0,66
0,0164
0,58
0,1661
ANGT_DPTFIPAPIQAK_vs_PGRP2_AGLLRPDYALLGHR
42 и 126
0,67
0,0074
0,54
0,5241
ANGT_DPTFIPAPIQAK_vs_TENX_LNWEAPPGAFDSFLLR
42 и 141
0,65
0,018
0,57
0,2186
APOC3_GWVTDGFSSLK_vs_IGF2_GIVEECCFR
47 и 103
0,59
0,1479
0,65
0,0087
APOC3_GWVTDGFSSLK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
47 и 135
0,57
0,2718
0,65
0,0081
APOH_ATVVYQGER_vs_IGF2_GIVEECCFR
48 и 103
0,56
0,3934
0,66
0,0056
B2MG_VEHSDLSFSK_vs_IGF2_GIVEECCFR
50 и 103
0,65
0,0222
0,57
0,2386
B2MG_VNHVTLSQPK_vs_IGF2_GIVEECCFR
51 и 103
0,66
0,0169
0,58
0,1465
BGH3_LTLLAPLNSVFK_vs_CHL1_VIAVNEVGR
52 и 66
0,65
0,0206
0,57
0,2597
BGH3_LTLLAPLNSVFK_vs_CRIS3_YEDLYSNCK
52 и 79
0,65
0,0262
0,6
0,0892
C1QB_VPGLYYFTYHASSR_vs_IGF2_GIVEECCFR
55 и 103
0,65
0,0226
0,62
0,0321
C1QB_VPGLYYFTYHASSR_vs_PGRP2_AGLLRPDYALLGHR
55 и 126
0,66
0,0164
0,58
0,1629
CD14_LTVGAAQVPAQLLVGALR_vs_IGF2_GIVEECCFR
61 и 103
0,65
0,0247
0,57
0,2206
CFAB_YGLVTYATYPK_vs_CHL1_VIAVNEVGR
64 и 66
0,66
0,014
0,57
0,2284
CFAB_YGLVTYATYPK_vs_CRIS3_YEDLYSNCK
64 и 79
0,65
0,0226
0,59
0,1395
CFAB_YGLVTYATYPK_vs_IBP3_YGQPLPGYTTK
64 и 100
0,65
0,0236
0,59
0,1381
CFAB_YGLVTYATYPK_vs_IGF2_GIVEECCFR
64 и 103
0,65
0,0209
0,62
0,0435
CFAB_YGLVTYATYPK_vs_LYAM1_SYYWIGIR
64 и 120
0,65
0,0222
0,61
0,0601
CFAB_YGLVTYATYPK_vs_PGRP2_AGLLRPDYALLGHR
64 и 126
0,67
0,0108
0,56
0,3133
CFAB_YGLVTYATYPK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
64 и 144
0,66
0,0166
0,6
0,0902
CO5_TLLPVSKPEIR_vs_PGRP2_AGLLRPDYALLGHR
70 и 126
0,66
0,0123
0,57
0,2206
CO5_TLLPVSKPEIR_vs_SHBG_IALGGLLFPASNLR
70 и 18
0,56
0,3419
0,65
0,0106
CO5_VFQFLEK_vs_PGRP2_AGLLRPDYALLGHR
71 и 126
0,66
0,0159
0,56
0,3387
CO6_ALNHLPLEYNSALYSR_vs_CHL1_VIAVNEVGR
72 и 66
0,65
0,0209
0,56
0,2753
ENPP2_TEFLSNYLTNVDDITLVPGTLGR_vs_IGF2_GIVEECCFR
82 и 103
0,65
0,0216
0,56
0,3083
FETUA_FSVVYAK_vs_IGF2_GIVEECCFR
88 и 103
0,65
0,0229
0,62
0,0429
HABP2_FLNWIK_vs_CHL1_VIAVNEVGR
92 и 66
0,65
0,02
0,56
0,3011
HABP2_FLNWIK_vs_IBP3_YGQPLPGYTTK
92 и 100
0,65
0,0229
0,59
0,1104
HABP2_FLNWIK_vs_IGF2_GIVEECCFR
92 и 103
0,65
0,0254
0,62
0,0321
IBP4_QCHPALDGQR_vs_CHL1_VIAVNEVGR
2 и 66
0,66
0,014
0,57
0,2597
IBP4_QCHPALDGQR_vs_IGF2_GIVEECCFR
2 и 103
0,66
0,0159
0,64
0,018
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,61
0,0895
0,65
0,0073
IBP4_QCHPALDGQR_vs_PGRP2_AGLLRPDYALLGHR
2 и 126
0,66
0,0149
0,6
0,0942
IBP4_QCHPALDGQR_vs_PSG3_VSAPSGTGHLPGLNPL
2 и 134
0,57
0,2572
0,66
0,0058
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,58
0,2385
0,67
0,0024
INHBC_LDFHFSSDR_vs_ALS_IRPHTFTGLSGLR
107 и 40
0,51
0,8798
0,72
2,00E-04
INHBC_LDFHFSSDR_vs_CRIS3_AVSPPAR
107 и 78
0,55
0,4808
0,66
0,0068
INHBC_LDFHFSSDR_vs_CRIS3_YEDLYSNCK
107 и 79
0,57
0,2644
0,65
0,0082
INHBC_LDFHFSSDR_vs_IBP3_FLNVLSPR
107 и 99
0,56
0,3903
0,7
6,00E-04
INHBC_LDFHFSSDR_vs_IBP3_YGQPLPGYTTK
107 и 100
0,57
0,2718
0,7
5,00E-04
INHBC_LDFHFSSDR_vs_IGF2_GIVEECCFR
107 и 103
0,57
0,2768
0,71
2,00E-04
INHBC_LDFHFSSDR_vs_ITIH4_ILDDLSPR
107 и 112
0,51
0,9204
0,67
0,0028
INHBC_LDFHFSSDR_vs_LYAM1_SYYWIGIR
107 и 120
0,54
0,5585
0,67
0,004
INHBC_LDFHFSSDR_vs_PGRP2_AGLLRPDYALLGHR
107 и 126
0,57
0,3082
0,65
0,0073
INHBC_LDFHFSSDR_vs_PSG3_VSAPSGTGHLPGLNPL
107 и 134
0,53
0,6459
0,72
1,00E-04
INHBC_LDFHFSSDR_vs_SHBG_IALGGLLFPASNLR
107 и 18
0,51
0,8439
0,7
6,00E-04
INHBC_LDFHFSSDR_vs_SPRL1_VLTHSELAPLR
107 и 140
0,56
0,3419
0,66
0,0062
INHBC_LDFHFSSDR_vs_TENX_LNWEAPPGAFDSFLLR
107 и 141
0,59
0,1479
0,67
0,0027
INHBC_LDFHFSSDR_vs_TENX_LSQLSVTDVTTSSLR
107 и 142
0,56
0,3477
0,66
0,0061
INHBC_LDFHFSSDR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
107 и 144
0,55
0,4632
0,67
0,004
INHBC_LDFHFSSDR_vs_VTDB_ELPEHTVK
107 и 147
0,52
0,7688
0,71
2,00E-04
ITIH3_ALDLSLK_vs_CRIS3_YEDLYSNCK
111 и 79
0,65
0,024
0,59
0,1116
ITIH3_ALDLSLK_vs_SHBG_IALGGLLFPASNLR
111 и 18
0,57
0,3082
0,66
0,0044
LBP_ITGFLKPGK_vs_CRIS3_AVSPPAR
118 и 78
0,65
0,0209
0,56
0,2708
LBP_ITGFLKPGK_vs_CRIS3_YEDLYSNCK
118 и 79
0,67
0,0089
0,57
0,2469
LBP_ITGFLKPGK_vs_LYAM1_SYYWIGIR
118 и 120
0,65
0,0216
0,57
0,1963
LBP_ITLPDFTGDLR_vs_CRIS3_AVSPPAR
119 и 78
0,68
0,005
0,58
0,1523
LBP_ITLPDFTGDLR_vs_CRIS3_YEDLYSNCK
119 и 79
0,7
0,0026
0,59
0,1327
LBP_ITLPDFTGDLR_vs_IBP3_YGQPLPGYTTK
119 и 100
0,69
0,0044
0,56
0,3387
LBP_ITLPDFTGDLR_vs_IGF2_GIVEECCFR
119 и 103
0,7
0,0024
0,57
0,2148
LBP_ITLPDFTGDLR_vs_LYAM1_SYYWIGIR
119 и 120
0,69
0,0038
0,6
0,0882
LBP_ITLPDFTGDLR_vs_PGRP2_AGLLRPDYALLGHR
119 и 126
0,69
0,0029
0,56
0,3133
LBP_ITLPDFTGDLR_vs_PSG3_VSAPSGTGHLPGLNPL
119 и 134
0,65
0,0197
0,6
0,0932
LBP_ITLPDFTGDLR_vs_TENX_LNWEAPPGAFDSFLLR
119 и 141
0,66
0,0121
0,56
0,2986
LBP_ITLPDFTGDLR_vs_VTDB_ELPEHTVK
119 и 147
0,68
0,0059
0,56
0,3059
PEDF_LQSLFDSPDFSK_vs_CRIS3_YEDLYSNCK
124 и 79
0,65
0,0229
0,61
0,0497
PSG2_IHPSYTNYR_vs_CRIS3_YEDLYSNCK
133 и 79
0,65
0,0229
0,56
0,2708
VTNC_GQYCYELDEK_vs_ALS_IRPHTFTGLSGLR
149 и 40
0,55
0,4844
0,67
0,0035
VTNC_GQYCYELDEK_vs_IBP3_YGQPLPGYTTK
149 и 100
0,65
0,0251
0,66
0,0071
VTNC_GQYCYELDEK_vs_IGF2_GIVEECCFR
149 и 103
0,63
0,0519
0,68
0,0023
VTNC_GQYCYELDEK_vs_ITIH4_ILDDLSPR
149 и 112
0,55
0,4325
0,65
0,0086
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,63
0,0407
0,66
0,0052
VTNC_GQYCYELDEK_vs_NCAM1_GLGEISAASEFK
149 и 121
0,53
0,6174
0,65
0,0082
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,65
0,0247
0,62
0,0369
VTNC_GQYCYELDEK_vs_PSG3_VSAPSGTGHLPGLNPL
149 и 134
0,57
0,2596
0,69
0,0012
VTNC_GQYCYELDEK_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
149 и 135
0,55
0,4359
0,66
0,0051
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,58
0,2273
0,69
0,0013
VTNC_GQYCYELDEK_vs_VTDB_ELPEHTVK
149 и 147
0,6
0,1311
0,68
0,0017
VTNC_VDTVDPPYPR_vs_CHL1_VIAVNEVGR
150 и 66
0,65
0,0226
0,59
0,1211
VTNC_VDTVDPPYPR_vs_IGF2_GIVEECCFR
150 и 103
0,62
0,0555
0,66
0,0065
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,63
0,0396
0,66
0,0063
VTNC_VDTVDPPYPR_vs_NCAM1_GLGEISAASEFK
150 и 121
0,54
0,5662
0,66
0,0068
VTNC_VDTVDPPYPR_vs_PSG3_VSAPSGTGHLPGLNPL
150 и 134
0,57
0,3028
0,67
0,0027
VTNC_VDTVDPPYPR_vs_PSG9_DVLLLVHNLPQNLPGYFWYK
150 и 135
0,54
0,5282
0,67
0,0034
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,58
0,204
0,68
0,0023
VTNC_VDTVDPPYPR_vs_VTDB_ELPEHTVK
150 и 147
0,61
0,0832
0,68
0,0015
Таблица 71: эффективность классификации по обращениям, недели 17-21.
AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, без BMI-стратификации, отдельно по полу плода.
Обращение
SEQ ID №
119_153_aBMI_35 жен. ROC_AUC
119_153_aBMI_35 жен. P-значение
119_153_aBMI_35 муж. ROC_AUC
119_153_aBMI_35 муж. P-значение
A2GL_DLLLPQPDLR_vs_LYAM1_SYYWIGIR
34 и 120
0,57
0,4262
0,67
0,0243
AFAM_DADPDTFFAK_vs_LYAM1_SYYWIGIR
37 и 120
0,51
0,9381
0,73
0,0018
AFAM_DADPDTFFAK_vs_PGRP2_AGLLRPDYALLGHR
37 и 126
0,51
0,9466
0,69
0,0116
AFAM_DADPDTFFAK_vs_SHBG_IALGGLLFPASNLR
37 и 18
0,53
0,7662
0,66
0,0248
AFAM_HFQNLGK_vs_IBP3_YGQPLPGYTTK
38 и 100
0,7
0,0336
0,62
0,1104
AFAM_HFQNLGK_vs_IGF2_GIVEECCFR
38 и 103
0,71
0,0263
0,63
0,0778
AFAM_HFQNLGK_vs_LYAM1_SYYWIGIR
38 и 120
0,51
0,921
0,73
0,0018
AFAM_HFQNLGK_vs_PGRP2_AGLLRPDYALLGHR
38 и 126
0,51
0,9594
0,69
0,0098
ANGT_DPTFIPAPIQAK_vs_LYAM1_SYYWIGIR
42 и 120
0,54
0,643
0,71
0,0035
ANGT_DPTFIPAPIQAK_vs_SHBG_IALGGLLFPASNLR
42 и 18
0,55
0,6315
0,67
0,0217
APOC3_GWVTDGFSSLK_vs_LYAM1_SYYWIGIR
47 и 120
0,57
0,4262
0,68
0,0146
APOH_ATVVYQGER_vs_CRIS3_YEDLYSNCK
48 и 79
0,6
0,2924
0,65
0,0451
APOH_ATVVYQGER_vs_LYAM1_SYYWIGIR
48 и 120
0,53
0,7377
0,71
0,0037
APOH_ATVVYQGER_vs_PGRP2_AGLLRPDYALLGHR
48 и 126
0,52
0,8198
0,69
0,0097
B2MG_VEHSDLSFSK_vs_LYAM1_SYYWIGIR
50 и 120
0,6
0,285
0,68
0,0166
B2MG_VNHVTLSQPK_vs_IGF2_GIVEECCFR
51 и 103
0,72
0,0217
0,56
0,4027
B2MG_VNHVTLSQPK_vs_LYAM1_SYYWIGIR
51 и 120
0,62
0,1848
0,71
0,0044
CATD_VGFAEAAR_vs_C163A_INPASLDK
57 и 54
0,63
0,176
0,73
0,0014
CATD_VGFAEAAR_vs_CRIS3_AVSPPAR
57 и 78
0,57
0,4387
0,68
0,0125
CATD_VGFAEAAR_vs_CRIS3_YEDLYSNCK
57 и 79
0,59
0,3124
0,68
0,0119
CATD_VGFAEAAR_vs_CSH_AHQLAIDTYQEFEETYIPK
57 и 80
0,63
0,183
0,68
0,0138
CATD_VGFAEAAR_vs_CSH_ISLLLIESWLEPVR
57 и 81
0,59
0,3443
0,69
0,0095
CATD_VGFAEAAR_vs_FBLN1_TGYYFDGISR
57 и 86
0,57
0,4547
0,68
0,0157
CATD_VGFAEAAR_vs_IBP1_VVESLAK
57 и 97
0,61
0,2593
0,67
0,02
CATD_VGFAEAAR_vs_IBP2_LIQGAPTIR
57 и 98
0,52
0,8198
0,67
0,0205
CATD_VGFAEAAR_vs_IGF2_GIVEECCFR
57 и 103
0,66
0,0821
0,67
0,0224
CATD_VGFAEAAR_vs_ITIH4_ILDDLSPR
57 и 112
0,54
0,6623
0,69
0,0111
CATD_VGFAEAAR_vs_LYAM1_SYYWIGIR
57 и 120
0,57
0,4324
0,76
4,00E-04
CATD_VGFAEAAR_vs_PGRP2_AGLLRPDYALLGHR
57 и 126
0,53
0,7621
0,72
0,0029
CATD_VGFAEAAR_vs_PSG3_VSAPSGTGHLPGLNPL
57 и 134
0,57
0,4678
0,7
0,0055
CATD_VGFAEAAR_vs_SHBG_IALGGLLFPASNLR
57 и 18
0,56
0,5538
0,69
0,0087
CATD_VGFAEAAR_vs_SOM2.CSH_NYGLLYCFR
57 и 138
0,61
0,2593
0,68
0,0139
CATD_VGFAEAAR_vs_SOM2.CSH_SVEGSCGF
57 и 139
0,58
0,381
0,71
0,0051
CATD_VGFAEAAR_vs_SPRL1_VLTHSELAPLR
57 и 140
0,6
0,2826
0,68
0,0125
CATD_VGFAEAAR_vs_TENX_LNWEAPPGAFDSFLLR
57 и 141
0,59
0,3202
0,71
0,0051
CATD_VGFAEAAR_vs_TENX_LSQLSVTDVTTSSLR
57 и 142
0,59
0,347
0,67
0,0181
CATD_VGFAEAAR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
57 и 144
0,59
0,3124
0,67
0,0207
CATD_VGFAEAAR_vs_VTDB_ELPEHTVK
57 и 147
0,56
0,5395
0,67
0,0187
CATD_VSTLPAITLK_vs_C163A_INPASLDK
58 и 54
0,58
0,3987
0,71
0,004
CATD_VSTLPAITLK_vs_CRIS3_AVSPPAR
58 и 78
0,56
0,4911
0,7
0,0071
CATD_VSTLPAITLK_vs_CRIS3_YEDLYSNCK
58 и 79
0,59
0,3497
0,7
0,0064
CATD_VSTLPAITLK_vs_CSH_AHQLAIDTYQEFEETYIPK
58 и 80
0,59
0,3308
0,66
0,0274
CATD_VSTLPAITLK_vs_CSH_ISLLLIESWLEPVR
58 и 81
0,57
0,458
0,66
0,0259
CATD_VSTLPAITLK_vs_FBLN1_TGYYFDGISR
58 и 86
0,57
0,4645
0,68
0,0131
CATD_VSTLPAITLK_vs_IBP1_VVESLAK
58 и 97
0,58
0,3957
0,67
0,0181
CATD_VSTLPAITLK_vs_IGF2_GIVEECCFR
58 и 103
0,66
0,0802
0,67
0,0203
CATD_VSTLPAITLK_vs_ITIH4_ILDDLSPR
58 и 112
0,52
0,8575
0,68
0,0152
CATD_VSTLPAITLK_vs_LYAM1_SYYWIGIR
58 и 120
0,55
0,6088
0,77
2,00E-04
CATD_VSTLPAITLK_vs_PGRP2_AGLLRPDYALLGHR
58 и 126
0,5
0,9679
0,74
0,0013
CATD_VSTLPAITLK_vs_PSG3_VSAPSGTGHLPGLNPL
58 и 134
0,58
0,4138
0,71
0,0043
CATD_VSTLPAITLK_vs_SHBG_IALGGLLFPASNLR
58 и 18
0,53
0,7296
0,7
0,0052
CATD_VSTLPAITLK_vs_SOM2.CSH_NYGLLYCFR
58 и 138
0,58
0,4077
0,66
0,0299
CATD_VSTLPAITLK_vs_SOM2.CSH_SVEGSCGF
58 и 139
0,55
0,5755
0,69
0,0116
CATD_VSTLPAITLK_vs_SPRL1_VLTHSELAPLR
58 и 140
0,57
0,4645
0,68
0,0136
CATD_VSTLPAITLK_vs_TENX_LNWEAPPGAFDSFLLR
58 и 141
0,58
0,3927
0,72
0,0032
CATD_VSTLPAITLK_vs_TENX_LSQLSVTDVTTSSLR
58 и 142
0,57
0,458
0,68
0,0141
CATD_VSTLPAITLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
58 и 144
0,56
0,536
0,67
0,0235
CATD_VSTLPAITLK_vs_VTDB_ELPEHTVK
58 и 147
0,54
0,6976
0,67
0,0187
CBPN_EALIQFLEQVHQGIK_vs_LYAM1_SYYWIGIR
59 и 120
0,61
0,2395
0,66
0,0302
CBPN_NGVDLNR_vs_LYAM1_SYYWIGIR
157 и 120
0,63
0,1693
0,68
0,0159
CD14_LTVGAAQVPAQLLVGALR_vs_IGF2_GIVEECCFR
61 и 103
0,72
0,0193
0,6
0,1921
CD14_LTVGAAQVPAQLLVGALR_vs_LYAM1_SYYWIGIR
61 и 120
0,61
0,2526
0,73
0,0014
CD14_LTVGAAQVPAQLLVGALR_vs_PGRP2_AGLLRPDYALLGHR
61 и 126
0,57
0,4711
0,68
0,0139
CD14_LTVGAAQVPAQLLVGALR_vs_SHBG_IALGGLLFPASNLR
61 и 18
0,58
0,3898
0,68
0,0153
CD14_LTVGAAQVPAQLLVGALR_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
61 и 144
0,69
0,0425
0,6
0,1631
CD14_LTVGAAQVPAQLLVGALR_vs_VTDB_ELPEHTVK
61 и 147
0,64
0,1428
0,67
0,0179
CD14_SWLAELQQWLKPGLK_vs_IGF2_GIVEECCFR
62 и 103
0,72
0,0175
0,57
0,3174
CD14_SWLAELQQWLKPGLK_vs_LYAM1_SYYWIGIR
62 и 120
0,62
0,1901
0,7
0,0059
CD14_SWLAELQQWLKPGLK_vs_PGRP2_AGLLRPDYALLGHR
62 и 126
0,57
0,4387
0,66
0,0306
CD14_SWLAELQQWLKPGLK_vs_SHBG_IALGGLLFPASNLR
62 и 18
0,58
0,3898
0,65
0,0351
CD14_SWLAELQQWLKPGLK_vs_TIE1_VSWSLPLVPGPLVGDGFLLR
62 и 144
0,71
0,029
0,59
0,204
CD14_SWLAELQQWLKPGLK_vs_VTDB_ELPEHTVK
62 и 147
0,66
0,0961
0,65
0,0442
CFAB_YGLVTYATYPK_vs_LYAM1_SYYWIGIR
64 и 120
0,52
0,866
0,67
0,0187
CLUS_ASSIIDELFQDR_vs_CRIS3_YEDLYSNCK
67 и 79
0,63
0,176
0,65
0,0465
CLUS_ASSIIDELFQDR_vs_IBP3_YGQPLPGYTTK
67 и 100
0,71
0,0267
0,56
0,4076
CLUS_ASSIIDELFQDR_vs_IGF2_GIVEECCFR
67 и 103
0,7
0,031
0,61
0,1288
CLUS_ASSIIDELFQDR_vs_LYAM1_SYYWIGIR
67 и 120
0,59
0,3334
0,73
0,0014
CLUS_ASSIIDELFQDR_vs_PGRP2_AGLLRPDYALLGHR
67 и 126
0,54
0,6937
0,67
0,0189
CLUS_ASSIIDELFQDR_vs_SHBG_IALGGLLFPASNLR
67 и 18
0,58
0,4017
0,67
0,0172
CLUS_ASSIIDELFQDR_vs_VTDB_ELPEHTVK
67 и 147
0,6
0,2779
0,68
0,0148
CLUS_LFDSDPITVTVPVEVSR_vs_CRIS3_YEDLYSNCK
68 и 79
0,63
0,1627
0,65
0,0446
CLUS_LFDSDPITVTVPVEVSR_vs_IBP3_YGQPLPGYTTK
68 и 100
0,7
0,0359
0,56
0,3861
CLUS_LFDSDPITVTVPVEVSR_vs_IGF2_GIVEECCFR
68 и 103
0,71
0,0267
0,6
0,1593
CLUS_LFDSDPITVTVPVEVSR_vs_LYAM1_SYYWIGIR
68 и 120
0,58
0,3898
0,72
0,0027
CLUS_LFDSDPITVTVPVEVSR_vs_PGRP2_AGLLRPDYALLGHR
68 и 126
0,53
0,7867
0,66
0,0326
CLUS_LFDSDPITVTVPVEVSR_vs_SHBG_IALGGLLFPASNLR
68 и 18
0,57
0,4324
0,66
0,0254
CLUS_LFDSDPITVTVPVEVSR_vs_VTDB_ELPEHTVK
68 и 147
0,58
0,3839
0,68
0,0146
CO5_TLLPVSKPEIR_vs_LYAM1_SYYWIGIR
70 и 120
0,61
0,2593
0,7
0,0056
CO5_VFQFLEK_vs_LYAM1_SYYWIGIR
71 и 120
0,53
0,7498
0,71
0,0043
CO6_ALNHLPLEYNSALYSR_vs_LYAM1_SYYWIGIR
72 и 120
0,58
0,4077
0,71
0,0049
CO8A_SLLQPNK_vs_IBP1_VVESLAK
74 и 97
0,57
0,4844
0,65
0,0398
CO8A_SLLQPNK_vs_LYAM1_SYYWIGIR
74 и 120
0,51
0,9551
0,74
0,001
CO8B_QALEEFQK_vs_IBP1_VVESLAK
76 и 97
0,55
0,5939
0,67
0,024
CO8B_QALEEFQK_vs_LYAM1_SYYWIGIR
76 и 120
0,55
0,5939
0,75
7,00E-04
CO8B_QALEEFQK_vs_PSG3_VSAPSGTGHLPGLNPL
76 и 134
0,51
0,8828
0,68
0,0161
F13B_GDTYPAELYITGSILR_vs_IBP3_YGQPLPGYTTK
84 и 100
0,74
0,0113
0,54
0,5451
F13B_GDTYPAELYITGSILR_vs_IGF2_GIVEECCFR
84 и 103
0,74
0,011
0,59
0,2071
F13B_GDTYPAELYITGSILR_vs_LYAM1_SYYWIGIR
84 и 120
0,63
0,1812
0,74
0,0011
F13B_GDTYPAELYITGSILR_vs_PGRP2_AGLLRPDYALLGHR
84 и 126
0,54
0,7016
0,67
0,021
F13B_GDTYPAELYITGSILR_vs_SHBG_IALGGLLFPASNLR
84 и 18
0,59
0,3281
0,66
0,0262
FETUA_FSVVYAK_vs_LYAM1_SYYWIGIR
88 и 120
0,56
0,5324
0,71
0,0037
FETUA_HTLNQIDEVK_vs_LYAM1_SYYWIGIR
89 и 120
0,51
0,9295
0,72
0,0032
HABP2_FLNWIK_vs_LYAM1_SYYWIGIR
92 и 120
0,54
0,6858
0,68
0,0161
HEMO_NFPSPVDAAFR_vs_LYAM1_SYYWIGIR
93 и 120
0,5
1
0,71
0,0037
IBP4_QCHPALDGQR_vs_CRIS3_AVSPPAR
2 и 78
0,58
0,3898
0,66
0,0248
IBP4_QCHPALDGQR_vs_CRIS3_YEDLYSNCK
2 и 79
0,61
0,229
0,67
0,0219
IBP4_QCHPALDGQR_vs_IGF2_GIVEECCFR
2 и 103
0,68
0,0541
0,57
0,33
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,54
0,6353
0,74
0,0013
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,53
0,7498
0,67
0,0229
IBP6_HLDSVLQQLQTEVYR_vs_LYAM1_SYYWIGIR
102 и 120
0,55
0,5755
0,68
0,0122
KNG1_DIPTNSPELEETLTHTITK_vs_LYAM1_SYYWIGIR
116 и 120
0,51
0,9423
0,73
0,002
KNG1_QVVAGLNFR_vs_CRIS3_AVSPPAR
117 и 78
0,57
0,4515
0,65
0,042
KNG1_QVVAGLNFR_vs_CRIS3_YEDLYSNCK
117 и 79
0,61
0,229
0,65
0,0428
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,58
0,4169
0,75
7,00E-04
KNG1_QVVAGLNFR_vs_PGRP2_AGLLRPDYALLGHR
117 и 126
0,54
0,6468
0,67
0,0212
KNG1_QVVAGLNFR_vs_SHBG_IALGGLLFPASNLR
117 и 18
0,6
0,285
0,66
0,0248
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,6
0,2899
0,71
0,0044
PEDF_TVQAVLTVPK_vs_IGF2_GIVEECCFR
125 и 103
0,7
0,0294
0,58
0,2682
PEDF_TVQAVLTVPK_vs_LYAM1_SYYWIGIR
125 и 120
0,59
0,3361
0,73
0,0015
PSG2_IHPSYTNYR_vs_CRIS3_AVSPPAR
133 и 78
0,61
0,2395
0,66
0,0326
PSG2_IHPSYTNYR_vs_CRIS3_YEDLYSNCK
133 и 79
0,63
0,1611
0,66
0,0316
PSG2_IHPSYTNYR_vs_FBLN1_TGYYFDGISR
133 и 86
0,57
0,4645
0,67
0,0189
PSG2_IHPSYTNYR_vs_IBP2_LIQGAPTIR
133 и 98
0,57
0,4483
0,66
0,0316
PSG2_IHPSYTNYR_vs_LYAM1_SYYWIGIR
133 и 120
0,59
0,3415
0,7
0,0072
PSG2_IHPSYTNYR_vs_PSG3_VSAPSGTGHLPGLNPL
133 и 134
0,6
0,3099
0,65
0,0363
PSG2_IHPSYTNYR_vs_SHBG_IALGGLLFPASNLR
133 и 18
0,58
0,3723
0,67
0,0189
PTGDS_GPGEDFR_vs_LYAM1_SYYWIGIR
137 и 120
0,55
0,6051
0,69
0,0094
THBG_AVLHIGEK_vs_LYAM1_SYYWIGIR
143 и 120
0,56
0,5395
0,69
0,0091
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,53
0,7176
0,76
4,00E-04
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,53
0,7826
0,7
0,0069
VTNC_GQYCYELDEK_vs_PSG3_VSAPSGTGHLPGLNPL
149 и 134
0,53
0,7703
0,68
0,0127
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,53
0,7296
0,68
0,0159
VTNC_VDTVDPPYPR_vs_CRIS3_YEDLYSNCK
150 и 79
0,58
0,3752
0,65
0,0451
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,52
0,8407
0,75
6,00E-04
VTNC_VDTVDPPYPR_vs_SHBG_IALGGLLFPASNLR
150 и 18
0,52
0,8744
0,68
0,0134
Таблица 72: эффективность классификации по обращениям, недели 17-21.
AUROC обращения для недель беременности от 17 0/7 до 21 6/7 с использованием порога случай-контроль <35 0/7 в сравнении с≥35 0/7 недель, с BMI-стратификацией (>22≤37), отдельно по полу плода.
Обращение
SEQ ID №
119_153_rBMI_35 жен. ROC_AUC
119_153_rBMI_35 жен. P-значение
119_153_rBMI_35 муж. ROC_AUC
119_153_rBMI_35 муж. P-значение
A2GL_DLLLPQPDLR_vs_CRIS3_YEDLYSNCK
34 и 79
0,76
0,0345
0,59
0,3046
A2GL_DLLLPQPDLR_vs_LYAM1_SYYWIGIR
34 и 120
0,7
0,0972
0,68
0,0323
AFAM_DADPDTFFAK_vs_LYAM1_SYYWIGIR
37 и 120
0,53
0,828
0,76
0,0018
AFAM_DADPDTFFAK_vs_PGRP2_AGLLRPDYALLGHR
37 и 126
0,51
0,962
0,74
0,0044
AFAM_DADPDTFFAK_vs_SOM2.CSH_SVEGSCGF
37 и 139
0,55
0,6755
0,67
0,0422
AFAM_HFQNLGK_vs_ALS_IRPHTFTGLSGLR
38 и 40
0,61
0,3649
0,67
0,0493
AFAM_HFQNLGK_vs_LYAM1_SYYWIGIR
38 и 120
0,54
0,7626
0,76
0,0019
AFAM_HFQNLGK_vs_PGRP2_AGLLRPDYALLGHR
38 и 126
0,5
0,9789
0,74
0,004
AFAM_HFQNLGK_vs_SOM2.CSH_SVEGSCGF
38 и 139
0,57
0,5494
0,69
0,0278
ANGT_DPTFIPAPIQAK_vs_LYAM1_SYYWIGIR
42 и 120
0,59
0,4361
0,75
0,0034
ANGT_DPTFIPAPIQAK_vs_PGRP2_AGLLRPDYALLGHR
42 и 126
0,54
0,7626
0,69
0,0224
ANGT_DPTFIPAPIQAK_vs_SHBG_IALGGLLFPASNLR
42 и 18
0,55
0,6755
0,69
0,0257
APOH_ATVVYQGER_vs_LYAM1_SYYWIGIR
48 и 120
0,54
0,7226
0,73
0,0071
APOH_ATVVYQGER_vs_PGRP2_AGLLRPDYALLGHR
48 и 126
0,51
0,9535
0,73
0,0072
B2MG_VNHVTLSQPK_vs_CRIS3_AVSPPAR
51 и 78
0,74
0,0446
0,59
0,3046
B2MG_VNHVTLSQPK_vs_CRIS3_YEDLYSNCK
51 и 79
0,78
0,02
0,6
0,251
B2MG_VNHVTLSQPK_vs_LYAM1_SYYWIGIR
51 и 120
0,71
0,091
0,71
0,0134
CATD_VGFAEAAR_vs_C163A_INPASLDK
57 и 54
0,68
0,1393
0,73
0,006
CATD_VGFAEAAR_vs_CRIS3_AVSPPAR
57 и 78
0,64
0,2374
0,67
0,0476
CATD_VGFAEAAR_vs_CRIS3_YEDLYSNCK
57 и 79
0,67
0,1572
0,67
0,0412
CATD_VGFAEAAR_vs_CSH_ISLLLIESWLEPVR
57 и 81
0,64
0,2416
0,68
0,0294
CATD_VGFAEAAR_vs_ITIH4_ILDDLSPR
57 и 112
0,55
0,6833
0,68
0,0362
CATD_VGFAEAAR_vs_LYAM1_SYYWIGIR
57 и 120
0,61
0,3705
0,77
0,0013
CATD_VGFAEAAR_vs_PGRP2_AGLLRPDYALLGHR
57 и 126
0,58
0,5353
0,72
0,011
CATD_VGFAEAAR_vs_SHBG_IALGGLLFPASNLR
57 и 18
0,56
0,6147
0,68
0,0342
CATD_VGFAEAAR_vs_SOM2.CSH_NYGLLYCFR
57 и 138
0,66
0,1835
0,67
0,0493
CATD_VGFAEAAR_vs_SOM2.CSH_SVEGSCGF
57 и 139
0,67
0,1572
0,68
0,0371
CATD_VGFAEAAR_vs_SPRL1_VLTHSELAPLR
57 и 140
0,58
0,4876
0,67
0,0397
CATD_VGFAEAAR_vs_TENX_LNWEAPPGAFDSFLLR
57 и 141
0,53
0,8363
0,72
0,011
CATD_VSTLPAITLK_vs_C163A_INPASLDK
58 и 54
0,64
0,2416
0,72
0,0087
CATD_VSTLPAITLK_vs_CRIS3_AVSPPAR
58 и 78
0,65
0,2092
0,69
0,0233
CATD_VSTLPAITLK_vs_CRIS3_YEDLYSNCK
58 и 79
0,69
0,118
0,7
0,0179
CATD_VSTLPAITLK_vs_IGF2_GIVEECCFR
58 и 103
0,74
0,0469
0,67
0,0484
CATD_VSTLPAITLK_vs_ITIH4_ILDDLSPR
58 и 112
0,53
0,828
0,68
0,0362
CATD_VSTLPAITLK_vs_LYAM1_SYYWIGIR
58 и 120
0,59
0,481
0,79
5,00E-04
CATD_VSTLPAITLK_vs_PGRP2_AGLLRPDYALLGHR
58 и 126
0,57
0,578
0,74
0,0042
CATD_VSTLPAITLK_vs_PSG3_VSAPSGTGHLPGLNPL
58 и 134
0,55
0,7068
0,7
0,0175
CATD_VSTLPAITLK_vs_SHBG_IALGGLLFPASNLR
58 и 18
0,54
0,7226
0,71
0,0149
CATD_VSTLPAITLK_vs_TENX_LNWEAPPGAFDSFLLR
58 и 141
0,52
0,8529
0,74
0,0048
CBPN_EALIQFLEQVHQGIK_vs_LYAM1_SYYWIGIR
59 и 120
0,6
0,4115
0,7
0,0206
CBPN_NGVDLNR_vs_LYAM1_SYYWIGIR
157 и 120
0,63
0,2821
0,72
0,0087
CD14_LTVGAAQVPAQLLVGALR_vs_CRIS3_AVSPPAR
61 и 78
0,76
0,0319
0,57
0,3774
CD14_LTVGAAQVPAQLLVGALR_vs_CRIS3_YEDLYSNCK
61 и 79
0,77
0,0237
0,6
0,2605
CD14_LTVGAAQVPAQLLVGALR_vs_LYAM1_SYYWIGIR
61 и 120
0,71
0,091
0,73
0,0077
CD14_LTVGAAQVPAQLLVGALR_vs_SOM2.CSH_SVEGSCGF
61 и 139
0,75
0,0393
0,59
0,2954
CD14_SWLAELQQWLKPGLK_vs_LYAM1_SYYWIGIR
62 и 120
0,69
0,1156
0,68
0,0311
CLUS_ASSIIDELFQDR_vs_LYAM1_SYYWIGIR
67 и 120
0,6
0,4115
0,74
0,0038
CLUS_ASSIIDELFQDR_vs_PGRP2_AGLLRPDYALLGHR
67 и 126
0,54
0,7305
0,67
0,0435
CLUS_LFDSDPITVTVPVEVSR_vs_LYAM1_SYYWIGIR
68 и 120
0,57
0,5708
0,7
0,0168
CO5_TLLPVSKPEIR_vs_LYAM1_SYYWIGIR
70 и 120
0,63
0,2681
0,72
0,0101
CO5_VFQFLEK_vs_LYAM1_SYYWIGIR
71 и 120
0,54
0,7147
0,71
0,0128
CO6_ALNHLPLEYNSALYSR_vs_LYAM1_SYYWIGIR
72 и 120
0,68
0,1451
0,7
0,0211
CO8A_SLLQPNK_vs_LYAM1_SYYWIGIR
74 и 120
0,53
0,8363
0,74
0,0042
CO8B_QALEEFQK_vs_LYAM1_SYYWIGIR
76 и 120
0,54
0,7385
0,73
0,0068
F13B_GDTYPAELYITGSILR_vs_CRIS3_AVSPPAR
84 и 78
0,75
0,0364
0,63
0,125
F13B_GDTYPAELYITGSILR_vs_CRIS3_YEDLYSNCK
84 и 79
0,77
0,0264
0,63
0,1307
F13B_GDTYPAELYITGSILR_vs_IGF2_GIVEECCFR
84 и 103
0,77
0,0271
0,59
0,2905
F13B_GDTYPAELYITGSILR_vs_LYAM1_SYYWIGIR
84 и 120
0,68
0,1422
0,76
0,0022
F13B_GDTYPAELYITGSILR_vs_PGRP2_AGLLRPDYALLGHR
84 и 126
0,58
0,5146
0,68
0,0305
F13B_GDTYPAELYITGSILR_vs_SHBG_IALGGLLFPASNLR
84 и 18
0,6
0,4055
0,67
0,0493
FETUA_FSVVYAK_vs_LYAM1_SYYWIGIR
88 и 120
0,59
0,4614
0,71
0,0145
FETUA_HTLNQIDEVK_vs_LYAM1_SYYWIGIR
89 и 120
0,56
0,6448
0,73
0,0077
HEMO_NFPSPVDAAFR_vs_LYAM1_SYYWIGIR
93 и 120
0,51
0,9451
0,72
0,0099
IBP4_QCHPALDGQR_vs_LYAM1_SYYWIGIR
2 и 120
0,58
0,5146
0,76
0,0021
IBP4_QCHPALDGQR_vs_SHBG_IALGGLLFPASNLR
2 и 18
0,52
0,8612
0,67
0,0443
IBP6_HLDSVLQQLQTEVYR_vs_LYAM1_SYYWIGIR
102 и 120
0,61
0,3593
0,7
0,0158
ITIH3_ALDLSLK_vs_LYAM1_SYYWIGIR
111 и 120
0,63
0,2727
0,68
0,0342
KNG1_DIPTNSPELEETLTHTITK_vs_LYAM1_SYYWIGIR
116 и 120
0,55
0,6678
0,73
0,006
KNG1_QVVAGLNFR_vs_IGF2_GIVEECCFR
117 и 103
0,74
0,0435
0,61
0,2099
KNG1_QVVAGLNFR_vs_LYAM1_SYYWIGIR
117 и 120
0,62
0,3375
0,76
0,0023
KNG1_QVVAGLNFR_vs_PGRP2_AGLLRPDYALLGHR
117 и 126
0,54
0,7385
0,69
0,0237
PAPP1_DIPHWLNPTR_vs_CRIS3_AVSPPAR
122 и 78
0,68
0,1338
0,67
0,0443
PAPP1_DIPHWLNPTR_vs_CRIS3_YEDLYSNCK
122 и 79
0,71
0,0795
0,67
0,0467
PAPP1_DIPHWLNPTR_vs_LYAM1_SYYWIGIR
122 и 120
0,64
0,2459
0,72
0,0106
PAPP1_DIPHWLNPTR_vs_PRG2_WNFAYWAAHQPWSR
122 и 129
0,76
0,0319
0,62
0,1714
PAPP1_DIPHWLNPTR_vs_SOM2.CSH_SVEGSCGF
122 и 139
0,72
0,0708
0,69
0,0268
PEDF_LQSLFDSPDFSK_vs_CRIS3_YEDLYSNCK
124 и 79
0,74
0,0493
0,61
0,1762
PEDF_LQSLFDSPDFSK_vs_LYAM1_SYYWIGIR
124 и 120
0,65
0,217
0,72
0,0079
PEDF_TVQAVLTVPK_vs_CRIS3_YEDLYSNCK
125 и 79
0,75
0,0373
0,62
0,1489
PEDF_TVQAVLTVPK_vs_LYAM1_SYYWIGIR
125 и 120
0,63
0,2774
0,76
0,0023
PEDF_TVQAVLTVPK_vs_PGRP2_AGLLRPDYALLGHR
125 и 126
0,57
0,5494
0,67
0,0451
PSG2_IHPSYTNYR_vs_CRIS3_AVSPPAR
133 и 78
0,76
0,0345
0,68
0,0355
PSG2_IHPSYTNYR_vs_CRIS3_YEDLYSNCK
133 и 79
0,77
0,0286
0,68
0,0369
PSG2_IHPSYTNYR_vs_FBLN1_TGYYFDGISR
133 и 86
0,61
0,3649
0,67
0,0476
PSG2_IHPSYTNYR_vs_IBP2_LIQGAPTIR
133 и 98
0,69
0,118
0,68
0,039
PSG2_IHPSYTNYR_vs_IBP3_YGQPLPGYTTK
133 и 100
0,74
0,0446
0,62
0,1599
PSG2_IHPSYTNYR_vs_IGF2_GIVEECCFR
133 и 103
0,76
0,0336
0,63
0,1287
PSG2_IHPSYTNYR_vs_LYAM1_SYYWIGIR
133 и 120
0,72
0,0759
0,74
0,005
PSG2_IHPSYTNYR_vs_PGRP2_AGLLRPDYALLGHR
133 и 126
0,69
0,1156
0,68
0,03
PSG2_IHPSYTNYR_vs_PSG3_VSAPSGTGHLPGLNPL
133 и 134
0,67
0,1541
0,68
0,0362
PSG2_IHPSYTNYR_vs_SHBG_IALGGLLFPASNLR
133 и 18
0,68
0,1283
0,71
0,0149
PSG2_IHPSYTNYR_vs_SOM2.CSH_SVEGSCGF
133 и 139
0,76
0,0345
0,63
0,1122
PTGDS_GPGEDFR_vs_LYAM1_SYYWIGIR
137 и 120
0,59
0,4614
0,72
0,0099
THBG_AVLHIGEK_vs_LYAM1_SYYWIGIR
143 и 120
0,59
0,4361
0,7
0,0211
VTNC_GQYCYELDEK_vs_LYAM1_SYYWIGIR
149 и 120
0,53
0,8363
0,81
3,00E-04
VTNC_GQYCYELDEK_vs_PGRP2_AGLLRPDYALLGHR
149 и 126
0,54
0,7626
0,75
0,0037
VTNC_GQYCYELDEK_vs_SHBG_IALGGLLFPASNLR
149 и 18
0,51
0,9282
0,69
0,0219
VTNC_VDTVDPPYPR_vs_LYAM1_SYYWIGIR
150 и 120
0,51
0,9451
0,76
0,0023
Таблица 73: скрининг пар антител к SHBG

Таблица 74: наиболее эффективные пары антител к SHBG с дополнительными калибраторами

Таблица 75: скрининг антител к IGFBP-4
Таблица 76: эффективность классификации по перехода, недели 19-20.
AUROC переходов для недель беременности от 19 1/7 до 20 6/7 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, без BMI-стратификации, для PTL.
Переход
SEQ ID №
134_146_aBMI_37 PTL ROC_AUC
PSG3_VSAPSGTGHLPGLNPL
134
0,66
IGF2_GIVEECCFR
103
0,66
IBP4_QCHPALDGQR
2
0,64
IBP3_YGQPLPGYTTK
100
0,64
F13B_GDTYPAELYITGSILR
84
0,61
APOH_ATVVYQGER
48
0,6
IBP3_FLNVLSPR
99
0,6
Таблица 77: эффективность классификации по переходам, недели 19-20.
AUROC переходов для недель беременности от 19 1/7 до 20 6/7 с использованием порога случай-контроль <37 0/7 в сравнении с≥37 0/7 недель, без BMI-стратификации, для PPROM.
Анализируемые вещества
SEQ ID №
134_146_aBMI_37 ROC_AUC
APOC3_GWVTDGFSSLK
47
0,76
PEDF_TVQAVLTVPK
125
0,76
INHBC_LDFHFSSDR
107
0,76
IBP4_QCHPALDGQR
2
0,73
PEDF_LQSLFDSPDFSK
124
0,73
A1AT_LSITGTYDLK
33
0,73
KNG1_QVVAGLNFR
117
0,72
CD14_LTVGAAQVPAQLLVGALR
61
0,72
VTNC_VDTVDPPYPR
150
0,71
KNG1_DIPTNSPELEETLTHTITK
116
0,71
CD14_SWLAELQQWLKPGLK
62
0,71
CO8A_SLLQPNK
74
0,69
CATD_VGFAEAAR
57
0,69
SHBG_IALGGLLFPASNLR
18
0,69
CO5_VFQFLEK
71
0,69
FETUA_FSVVYAK
88
0,68
HABP2_FLNWIK
92
0,68
VTNC_GQYCYELDEK
149
0,68
B2MG_VNHVTLSQPK
51
0,68
ENPP2_TYLHTYESEI
83
0,67
AFAM_HFQNLGK
38
0,67
APOH_ATVVYQGER
48
0,66
ITIH4_NPLVWVHASPEHVVVTR
113
0,66
CFAB_YGLVTYATYPK
64
0,66
CO8B_QALEEFQK
76
0,65
BGH3_LTLLAPLNSVFK
52
0,65
FETUA_HTLNQIDEVK
89
0,65
CO3_IHWESASLLR
69
0,65
ENPP2_TEFLSNYLTNVDDITLVPGTLGR
82
0,65
HEMO_NFPSPVDAAFR
93
0,65
LBP_ITLPDFTGDLR
119
0,65
CO5_TLLPVSKPEIR
70
0,65
FIBA_ESSSHHPGIAEFPSR
90
0,64
AFAM_DADPDTFFAK
37
0,64
ITIH3_ALDLSLK
111
0,64
LBP_ITGFLKPGK
118
0,64
CATD_VSTLPAITLK
58
0,64
PRDX2_GLFIIDGK
128
0,63
CO6_ALNHLPLEYNSALYSR
72
0,63
ANGT_DPTFIPAPIQAK
42
0,62
PRG2_WNFAYWAAHQPWSR
129
0,62
CBPN_NNANGVDLNR
60
0,62
IBP6_HLDSVLQQLQTEVYR
102
0,62
ITIH4_ILDDLSPR
112
0,62
IBP6_GAQTLYVPNCDHR
101
0,62
F13B_GDTYPAELYITGSILR
84
0,61
CLUS_LFDSDPITVTVPVEVSR
68
0,61
FBLN1_TGYYFDGISR
86
0,61
PAPP1_DIPHWLNPTR
122
0,61
TIE1_VSWSLPLVPGPLVGDGFLLR
144
0,6
CAH1_GGPFSDSYR
56
0,6
A2GL_DLLLPQPDLR
34
0,6
B2MG_VEHSDLSFSK
50
0,6
PSG2_IHPSYTNYR
133
0,6
--->
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> SERA PROGNOSTICS, INC.
<120> ПАРЫ БИОЛОГИЧЕСКИХ МАРКЕРОВ ДЛЯ ПРЕДСКАЗАНИЯ ПРЕЖДЕВРЕМЕННЫХ РОДОВ
<130> 13271-018-228
<140> PCT/US2016/038198
<141> 2016-06-17
<150> 62/290,796
<151> 2016-02-03
<150> 62/387,420
<151> 2015-12-24
<150> 62/182,349
<151> 2015-06-19
<160> 161
<170> PatentIn version 3.5
<210> 1
<211> 8
<212> Белок
<213> Homo sapiens
<400> 1
Leu Pro Gly Gly Leu Glu Pro Lys
1 5
<210> 2
<211> 10
<212> Белок
<213> Homo sapiens
<400> 2
Gln Cys His Pro Ala Leu Asp Gly Gln Arg
1 5 10
<210> 3
<211> 15
<212> Белок
<213> Homo sapiens
<400> 3
Thr His Glu Asp Leu Tyr Ile Ile Pro Ile Pro Asn Cys Asp Arg
1 5 10 15
<210> 4
<211> 17
<212> Белок
<213> Homo sapiens
<400> 4
Glu Asp Ala Arg Pro Val Pro Gln Gly Ser Cys Gln Ser Glu Leu His
1 5 10 15
Arg
<210> 5
<211> 12
<212> Белок
<213> Homo sapiens
<400> 5
Cys Arg Pro Pro Val Gly Cys Glu Glu Leu Val Arg
1 5 10
<210> 6
<211> 6
<212> Белок
<213> Homo sapiens
<400> 6
Val Asn Gly Ala Pro Arg
1 5
<210> 7
<211> 7
<212> Белок
<213> Homo sapiens
<400> 7
Leu Ala Ala Ser Gln Ser Arg
1 5
<210> 8
<211> 7
<212> Белок
<213> Homo sapiens
<400> 8
Asn Gly Asn Phe His Pro Lys
1 5
<210> 9
<211> 6
<212> Белок
<213> Homo sapiens
<400> 9
Cys Trp Cys Val Asp Arg
1 5
<210> 10
<211> 13
<212> Белок
<213> Homo sapiens
<400> 10
Gly Glu Leu Asp Cys His Gln Leu Ala Asp Ser Phe Arg
1 5 10
<210> 11
<211> 8
<212> Белок
<213> Homo sapiens
<400> 11
Thr Ser Ser Ser Phe Glu Val Arg
1 5
<210> 12
<211> 16
<212> Белок
<213> Homo sapiens
<400> 12
Thr Trp Asp Pro Glu Gly Val Ile Phe Tyr Gly Asp Thr Asn Pro Lys
1 5 10 15
<210> 13
<211> 9
<212> Белок
<213> Homo sapiens
<400> 13
Asp Asp Trp Phe Met Leu Gly Leu Arg
1 5
<210> 14
<211> 22
<212> Белок
<213> Homo sapiens
<400> 14
Asp Gly Arg Pro Glu Ile Gln Leu His Asn His Trp Ala Gln Leu Thr
1 5 10 15
Val Gly Ala Gly Pro Arg
20
<210> 15
<211> 7
<212> Белок
<213> Homo sapiens
<400> 15
Trp His Gln Val Glu Val Lys
1 5
<210> 16
<211> 17
<212> Белок
<213> Homo sapiens
<400> 16
Met Glu Gly Asp Ser Val Leu Leu Glu Val Asp Gly Glu Glu Val Leu
1 5 10 15
Arg
<210> 17
<211> 9
<212> Белок
<213> Homo sapiens
<400> 17
Gln Val Ser Gly Pro Leu Thr Ser Lys
1 5
<210> 18
<211> 14
<212> Белок
<213> Homo sapiens
<400> 18
Ile Ala Leu Gly Gly Leu Leu Phe Pro Ala Ser Asn Leu Arg
1 5 10
<210> 19
<211> 12
<212> Белок
<213> Homo sapiens
<400> 19
Leu Pro Leu Val Pro Ala Leu Asp Gly Cys Leu Arg
1 5 10
<210> 20
<211> 6
<212> Белок
<213> Homo sapiens
<400> 20
Asp Ser Trp Leu Asp Lys
1 5
<210> 21
<211> 13
<212> Белок
<213> Homo sapiens
<400> 21
Gln Ala Glu Ile Ser Ala Ser Ala Pro Thr Ser Leu Arg
1 5 10
<210> 22
<211> 23
<212> Белок
<213> Homo sapiens
<400> 22
Ser Cys Asp Val Glu Ser Asn Pro Gly Ile Phe Leu Pro Pro Gly Thr
1 5 10 15
Gln Ala Glu Phe Asn Leu Arg
20
<210> 23
<211> 19
<212> Белок
<213> Homo sapiens
<400> 23
Asp Ile Pro Gln Pro His Ala Glu Pro Trp Ala Phe Ser Leu Asp Leu
1 5 10 15
Gly Leu Lys
<210> 24
<211> 24
<212> Белок
<213> Homo sapiens
<400> 24
Val Val Leu Ser Ser Gly Ser Gly Pro Gly Leu Asp Leu Pro Leu Val
1 5 10 15
Leu Gly Leu Pro Leu Gln Leu Lys
20
<210> 25
<211> 8
<212> Белок
<213> Homo sapiens
<400> 25
Val Val Leu Ser Gln Gly Ser Lys
1 5
<210> 26
<211> 9
<212> Белок
<213> Homo sapiens
<400> 26
Leu Asp Val Asp Gln Ala Leu Asn Arg
1 5
<210> 27
<211> 22
<212> Белок
<213> Homo sapiens
<400> 27
Ala Leu Ala Leu Pro Pro Leu Gly Leu Ala Pro Leu Leu Asn Leu Trp
1 5 10 15
Ala Lys Pro Gln Gly Arg
20
<210> 28
<211> 21
<212> Белок
<213> Homo sapiens
<400> 28
Ser His Glu Ile Trp Thr His Ser Cys Pro Gln Ser Pro Gly Asn Gly
1 5 10 15
Thr Asp Ala Ser His
20
<210> 29
<211> 14
<212> Белок
<213> Homo sapiens
<400> 29
Leu Pro Ala Glu Ile Ser Ala Ser Ala Pro Thr Ser Leu Arg
1 5 10
<210> 30
<211> 7
<212> Белок
<213> Homo sapiens
<400> 30
Thr Leu Pro Pro Leu Phe Ala
1 5
<210> 31
<211> 19
<212> Белок
<213> Homo sapiens
<400> 31
Gly Glu Asp Ser Ser Thr Ser Phe Cys Leu Asn Gly Leu Trp Ala Gln
1 5 10 15
Gly Gln Arg
<210> 32
<211> 15
<212> Белок
<213> Homo sapiens
<400> 32
Asn Trp Gly Leu Ser Val Tyr Ala Asp Lys Pro Glu Thr Thr Lys
1 5 10 15
<210> 33
<211> 10
<212> Белок
<213> Homo sapiens
<400> 33
Leu Ser Ile Thr Gly Thr Tyr Asp Leu Lys
1 5 10
<210> 34
<211> 10
<212> Белок
<213> Homo sapiens
<400> 34
Asp Leu Leu Leu Pro Gln Pro Asp Leu Arg
1 5 10
<210> 35
<211> 6
<212> Белок
<213> Homo sapiens
<400> 35
Leu Gln Val Leu Gly Lys
1 5
<210> 36
<211> 19
<212> Белок
<213> Homo sapiens
<400> 36
Leu His Thr Glu Ala Gln Ile Gln Glu Glu Gly Thr Val Val Glu Leu
1 5 10 15
Thr Gly Arg
<210> 37
<211> 10
<212> Белок
<213> Homo sapiens
<400> 37
Asp Ala Asp Pro Asp Thr Phe Phe Ala Lys
1 5 10
<210> 38
<211> 7
<212> Белок
<213> Homo sapiens
<400> 38
His Phe Gln Asn Leu Gly Lys
1 5
<210> 39
<211> 7
<212> Белок
<213> Homo sapiens
<400> 39
Leu Val Thr Asp Leu Thr Lys
1 5
<210> 40
<211> 13
<212> Белок
<213> Homo sapiens
<400> 40
Ile Arg Pro His Thr Phe Thr Gly Leu Ser Gly Leu Arg
1 5 10
<210> 41
<211> 8
<212> Белок
<213> Homo sapiens
<400> 41
Leu Glu Tyr Leu Leu Leu Ser Arg
1 5
<210> 42
<211> 12
<212> Белок
<213> Homo sapiens
<400> 42
Asp Pro Thr Phe Ile Pro Ala Pro Ile Gln Ala Lys
1 5 10
<210> 43
<211> 13
<212> Белок
<213> Homo sapiens
<400> 43
Ser Leu Asp Phe Thr Glu Leu Asp Val Ala Ala Glu Lys
1 5 10
<210> 44
<211> 9
<212> Белок
<213> Homo sapiens
<400> 44
Ala Lys Pro Ala Leu Glu Asp Leu Arg
1 5
<210> 45
<211> 9
<212> Белок
<213> Homo sapiens
<400> 45
Ser Pro Glu Leu Gln Ala Glu Ala Lys
1 5
<210> 46
<211> 7
<212> Белок
<213> Homo sapiens
<400> 46
Asp Tyr Trp Ser Thr Val Lys
1 5
<210> 47
<211> 11
<212> Белок
<213> Homo sapiens
<400> 47
Gly Trp Val Thr Asp Gly Phe Ser Ser Leu Lys
1 5 10
<210> 48
<211> 9
<212> Белок
<213> Homo sapiens
<400> 48
Ala Thr Val Val Tyr Gln Gly Glu Arg
1 5
<210> 49
<211> 9
<212> Белок
<213> Homo sapiens
<400> 49
Glu His Ser Ser Leu Ala Phe Trp Lys
1 5
<210> 50
<211> 10
<212> Белок
<213> Homo sapiens
<400> 50
Val Glu His Ser Asp Leu Ser Phe Ser Lys
1 5 10
<210> 51
<211> 10
<212> Белок
<213> Homo sapiens
<400> 51
Val Asn His Val Thr Leu Ser Gln Pro Lys
1 5 10
<210> 52
<211> 12
<212> Белок
<213> Homo sapiens
<400> 52
Leu Thr Leu Leu Ala Pro Leu Asn Ser Val Phe Lys
1 5 10
<210> 53
<211> 7
<212> Белок
<213> Homo sapiens
<400> 53
Val Leu Thr Asp Glu Leu Lys
1 5
<210> 54
<211> 8
<212> Белок
<213> Homo sapiens
<400> 54
Ile Asn Pro Ala Ser Leu Asp Lys
1 5
<210> 55
<211> 14
<212> Белок
<213> Homo sapiens
<400> 55
Val Pro Gly Leu Tyr Tyr Phe Thr Tyr His Ala Ser Ser Arg
1 5 10
<210> 56
<211> 9
<212> Белок
<213> Homo sapiens
<400> 56
Gly Gly Pro Phe Ser Asp Ser Tyr Arg
1 5
<210> 57
<211> 8
<212> Белок
<213> Homo sapiens
<400> 57
Val Gly Phe Ala Glu Ala Ala Arg
1 5
<210> 58
<211> 10
<212> Белок
<213> Homo sapiens
<400> 58
Val Ser Thr Leu Pro Ala Ile Thr Leu Lys
1 5 10
<210> 59
<211> 15
<212> Белок
<213> Homo sapiens
<400> 59
Glu Ala Leu Ile Gln Phe Leu Glu Gln Val His Gln Gly Ile Lys
1 5 10 15
<210> 60
<211> 10
<212> Белок
<213> Homo sapiens
<400> 60
Asn Asn Ala Asn Gly Val Asp Leu Asn Arg
1 5 10
<210> 61
<211> 18
<212> Белок
<213> Homo sapiens
<400> 61
Leu Thr Val Gly Ala Ala Gln Val Pro Ala Gln Leu Leu Val Gly Ala
1 5 10 15
Leu Arg
<210> 62
<211> 15
<212> Белок
<213> Homo sapiens
<400> 62
Ser Trp Leu Ala Glu Leu Gln Gln Trp Leu Lys Pro Gly Leu Lys
1 5 10 15
<210> 63
<211> 14
<212> Белок
<213> Homo sapiens
<400> 63
Val Ser Glu Ala Asp Ser Ser Asn Ala Asp Trp Val Thr Lys
1 5 10
<210> 64
<211> 11
<212> Белок
<213> Homo sapiens
<400> 64
Tyr Gly Leu Val Thr Tyr Ala Thr Tyr Pro Lys
1 5 10
<210> 65
<211> 10
<212> Белок
<213> Homo sapiens
<400> 65
Thr Ala Val Thr Ala Asn Leu Asp Ile Arg
1 5 10
<210> 66
<211> 9
<212> Белок
<213> Homo sapiens
<400> 66
Val Ile Ala Val Asn Glu Val Gly Arg
1 5
<210> 67
<211> 12
<212> Белок
<213> Homo sapiens
<400> 67
Ala Ser Ser Ile Ile Asp Glu Leu Phe Gln Asp Arg
1 5 10
<210> 68
<211> 17
<212> Белок
<213> Homo sapiens
<400> 68
Leu Phe Asp Ser Asp Pro Ile Thr Val Thr Val Pro Val Glu Val Ser
1 5 10 15
Arg
<210> 69
<211> 10
<212> Белок
<213> Homo sapiens
<400> 69
Ile His Trp Glu Ser Ala Ser Leu Leu Arg
1 5 10
<210> 70
<211> 11
<212> Белок
<213> Homo sapiens
<400> 70
Thr Leu Leu Pro Val Ser Lys Pro Glu Ile Arg
1 5 10
<210> 71
<211> 7
<212> Белок
<213> Homo sapiens
<400> 71
Val Phe Gln Phe Leu Glu Lys
1 5
<210> 72
<211> 16
<212> Белок
<213> Homo sapiens
<400> 72
Ala Leu Asn His Leu Pro Leu Glu Tyr Asn Ser Ala Leu Tyr Ser Arg
1 5 10 15
<210> 73
<211> 11
<212> Белок
<213> Homo sapiens
<400> 73
Ser Glu Tyr Gly Ala Ala Leu Ala Trp Glu Lys
1 5 10
<210> 74
<211> 7
<212> Белок
<213> Homo sapiens
<400> 74
Ser Leu Leu Gln Pro Asn Lys
1 5
<210> 75
<211> 25
<212> Белок
<213> Homo sapiens
<400> 75
Tyr His Phe Glu Ala Leu Ala Asp Thr Gly Ile Ser Ser Glu Phe Tyr
1 5 10 15
Asp Asn Ala Asn Asp Leu Leu Ser Lys
20 25
<210> 76
<211> 8
<212> Белок
<213> Homo sapiens
<400> 76
Gln Ala Leu Glu Glu Phe Gln Lys
1 5
<210> 77
<211> 8
<212> Белок
<213> Homo sapiens
<400> 77
Ser Gly Phe Ser Phe Gly Phe Lys
1 5
<210> 78
<211> 7
<212> Белок
<213> Homo sapiens
<400> 78
Ala Val Ser Pro Pro Ala Arg
1 5
<210> 79
<211> 9
<212> Белок
<213> Homo sapiens
<400> 79
Tyr Glu Asp Leu Tyr Ser Asn Cys Lys
1 5
<210> 80
<211> 19
<212> Белок
<213> Homo sapiens
<400> 80
Ala His Gln Leu Ala Ile Asp Thr Tyr Gln Glu Phe Glu Glu Thr Tyr
1 5 10 15
Ile Pro Lys
<210> 81
<211> 14
<212> Белок
<213> Homo sapiens
<400> 81
Ile Ser Leu Leu Leu Ile Glu Ser Trp Leu Glu Pro Val Arg
1 5 10
<210> 82
<211> 23
<212> Белок
<213> Homo sapiens
<400> 82
Thr Glu Phe Leu Ser Asn Tyr Leu Thr Asn Val Asp Asp Ile Thr Leu
1 5 10 15
Val Pro Gly Thr Leu Gly Arg
20
<210> 83
<211> 10
<212> Белок
<213> Homo sapiens
<400> 83
Thr Tyr Leu His Thr Tyr Glu Ser Glu Ile
1 5 10
<210> 84
<211> 16
<212> Белок
<213> Homo sapiens
<400> 84
Gly Asp Thr Tyr Pro Ala Glu Leu Tyr Ile Thr Gly Ser Ile Leu Arg
1 5 10 15
<210> 85
<211> 9
<212> Белок
<213> Homo sapiens
<400> 85
Ile Ala Gln Tyr Tyr Tyr Thr Phe Lys
1 5
<210> 86
<211> 10
<212> Белок
<213> Homo sapiens
<400> 86
Thr Gly Tyr Tyr Phe Asp Gly Ile Ser Arg
1 5 10
<210> 87
<211> 8
<212> Белок
<213> Homo sapiens
<400> 87
Ile Pro Ser Asn Pro Ser His Arg
1 5
<210> 88
<211> 7
<212> Белок
<213> Homo sapiens
<400> 88
Phe Ser Val Val Tyr Ala Lys
1 5
<210> 89
<211> 10
<212> Белок
<213> Homo sapiens
<400> 89
His Thr Leu Asn Gln Ile Asp Glu Val Lys
1 5 10
<210> 90
<211> 15
<212> Белок
<213> Homo sapiens
<400> 90
Glu Ser Ser Ser His His Pro Gly Ile Ala Glu Phe Pro Ser Arg
1 5 10 15
<210> 91
<211> 13
<212> Белок
<213> Homo sapiens
<400> 91
Gln Gly Phe Gly Asn Val Ala Thr Asn Thr Asp Gly Lys
1 5 10
<210> 92
<211> 6
<212> Белок
<213> Homo sapiens
<400> 92
Phe Leu Asn Trp Ile Lys
1 5
<210> 93
<211> 11
<212> Белок
<213> Homo sapiens
<400> 93
Asn Phe Pro Ser Pro Val Asp Ala Ala Phe Arg
1 5 10
<210> 94
<211> 16
<212> Белок
<213> Homo sapiens
<400> 94
Ser Gly Ala Gln Ala Thr Trp Thr Glu Leu Pro Trp Pro His Glu Lys
1 5 10 15
<210> 95
<211> 13
<212> Белок
<213> Homo sapiens
<400> 95
Trp Ala Ala Val Val Val Pro Ser Gly Glu Glu Gln Arg
1 5 10
<210> 96
<211> 13
<212> Белок
<213> Homo sapiens
<400> 96
Thr Glu Gly Asp Gly Val Tyr Thr Leu Asn Asn Glu Lys
1 5 10
<210> 97
<211> 7
<212> Белок
<213> Homo sapiens
<400> 97
Val Val Glu Ser Leu Ala Lys
1 5
<210> 98
<211> 9
<212> Белок
<213> Homo sapiens
<400> 98
Leu Ile Gln Gly Ala Pro Thr Ile Arg
1 5
<210> 99
<211> 8
<212> Белок
<213> Homo sapiens
<400> 99
Phe Leu Asn Val Leu Ser Pro Arg
1 5
<210> 100
<211> 11
<212> Белок
<213> Homo sapiens
<400> 100
Tyr Gly Gln Pro Leu Pro Gly Tyr Thr Thr Lys
1 5 10
<210> 101
<211> 13
<212> Белок
<213> Homo sapiens
<400> 101
Gly Ala Gln Thr Leu Tyr Val Pro Asn Cys Asp His Arg
1 5 10
<210> 102
<211> 15
<212> Белок
<213> Homo sapiens
<400> 102
His Leu Asp Ser Val Leu Gln Gln Leu Gln Thr Glu Val Tyr Arg
1 5 10 15
<210> 103
<211> 9
<212> Белок
<213> Homo sapiens
<400> 103
Gly Ile Val Glu Glu Cys Cys Phe Arg
1 5
<210> 104
<211> 16
<212> Белок
<213> Homo sapiens
<400> 104
Ser Cys Asp Leu Ala Leu Leu Glu Thr Tyr Cys Ala Thr Pro Ala Lys
1 5 10 15
<210> 105
<211> 8
<212> Белок
<213> Homo sapiens
<400> 105
Ala Leu Pro Ala Pro Ile Glu Lys
1 5
<210> 106
<211> 7
<212> Белок
<213> Homo sapiens
<400> 106
Gly Phe Pro Ser Val Leu Arg
1 5
<210> 107
<211> 9
<212> Белок
<213> Homo sapiens
<400> 107
Leu Asp Phe His Phe Ser Ser Asp Arg
1 5
<210> 108
<211> 7
<212> Белок
<213> Homo sapiens
<400> 108
Ala Ser Ser Ile Leu Ala Thr
1 5
<210> 109
<211> 12
<212> Белок
<213> Homo sapiens
<400> 109
Glu Leu Trp Phe Ser Asp Asp Pro Asp Val Thr Lys
1 5 10
<210> 110
<211> 11
<212> Белок
<213> Homo sapiens
<400> 110
Asn Val Asp Gln Ser Leu Leu Glu Leu His Lys
1 5 10
<210> 111
<211> 7
<212> Белок
<213> Homo sapiens
<400> 111
Ala Leu Asp Leu Ser Leu Lys
1 5
<210> 112
<211> 8
<212> Белок
<213> Homo sapiens
<400> 112
Ile Leu Asp Asp Leu Ser Pro Arg
1 5
<210> 113
<211> 17
<212> Белок
<213> Homo sapiens
<400> 113
Asn Pro Leu Val Trp Val His Ala Ser Pro Glu His Val Val Val Thr
1 5 10 15
Arg
<210> 114
<211> 19
<212> Белок
<213> Homo sapiens
<400> 114
Gln Leu Gly Leu Pro Gly Pro Pro Asp Val Pro Asp His Ala Ala Tyr
1 5 10 15
His Pro Phe
<210> 115
<211> 8
<212> Белок
<213> Homo sapiens
<400> 115
Val Arg Pro Gln Gln Leu Val Lys
1 5
<210> 116
<211> 19
<212> Белок
<213> Homo sapiens
<400> 116
Asp Ile Pro Thr Asn Ser Pro Glu Leu Glu Glu Thr Leu Thr His Thr
1 5 10 15
Ile Thr Lys
<210> 117
<211> 9
<212> Белок
<213> Homo sapiens
<400> 117
Gln Val Val Ala Gly Leu Asn Phe Arg
1 5
<210> 118
<211> 9
<212> Белок
<213> Homo sapiens
<400> 118
Ile Thr Gly Phe Leu Lys Pro Gly Lys
1 5
<210> 119
<211> 11
<212> Белок
<213> Homo sapiens
<400> 119
Ile Thr Leu Pro Asp Phe Thr Gly Asp Leu Arg
1 5 10
<210> 120
<211> 8
<212> Белок
<213> Homo sapiens
<400> 120
Ser Tyr Tyr Trp Ile Gly Ile Arg
1 5
<210> 121
<211> 12
<212> Белок
<213> Homo sapiens
<400> 121
Gly Leu Gly Glu Ile Ser Ala Ala Ser Glu Phe Lys
1 5 10
<210> 122
<211> 10
<212> Белок
<213> Homo sapiens
<400> 122
Asp Ile Pro His Trp Leu Asn Pro Thr Arg
1 5 10
<210> 123
<211> 13
<212> Белок
<213> Homo sapiens
<400> 123
Leu Asp Gly Ser Thr His Leu Asn Ile Phe Phe Ala Lys
1 5 10
<210> 124
<211> 12
<212> Белок
<213> Homo sapiens
<400> 124
Leu Gln Ser Leu Phe Asp Ser Pro Asp Phe Ser Lys
1 5 10
<210> 125
<211> 10
<212> Белок
<213> Homo sapiens
<400> 125
Thr Val Gln Ala Val Leu Thr Val Pro Lys
1 5 10
<210> 126
<211> 14
<212> Белок
<213> Homo sapiens
<400> 126
Ala Gly Leu Leu Arg Pro Asp Tyr Ala Leu Leu Gly His Arg
1 5 10
<210> 127
<211> 20
<212> Белок
<213> Homo sapiens
<400> 127
Asp Gly Ser Pro Asp Val Thr Thr Ala Asp Ile Gly Ala Asn Thr Pro
1 5 10 15
Asp Ala Thr Lys
20
<210> 128
<211> 8
<212> Белок
<213> Homo sapiens
<400> 128
Gly Leu Phe Ile Ile Asp Gly Lys
1 5
<210> 129
<211> 14
<212> Белок
<213> Homo sapiens
<400> 129
Trp Asn Phe Ala Tyr Trp Ala Ala His Gln Pro Trp Ser Arg
1 5 10
<210> 130
<211> 25
<212> Белок
<213> Homo sapiens
<400> 130
Asp Leu Tyr His Tyr Ile Thr Ser Tyr Val Val Asp Gly Glu Ile Ile
1 5 10 15
Ile Tyr Gly Pro Ala Tyr Ser Gly Arg
20 25
<210> 131
<211> 7
<212> Белок
<213> Homo sapiens
<400> 131
Phe Gln Leu Pro Gly Gln Lys
1 5
<210> 132
<211> 9
<212> Белок
<213> Homo sapiens
<400> 132
Leu Phe Ile Pro Gln Ile Thr Pro Lys
1 5
<210> 133
<211> 9
<212> Белок
<213> Homo sapiens
<400> 133
Ile His Pro Ser Tyr Thr Asn Tyr Arg
1 5
<210> 134
<211> 16
<212> Белок
<213> Homo sapiens
<400> 134
Val Ser Ala Pro Ser Gly Thr Gly His Leu Pro Gly Leu Asn Pro Leu
1 5 10 15
<210> 135
<211> 20
<212> Белок
<213> Homo sapiens
<400> 135
Asp Val Leu Leu Leu Val His Asn Leu Pro Gln Asn Leu Pro Gly Tyr
1 5 10 15
Phe Trp Tyr Lys
20
<210> 136
<211> 8
<212> Белок
<213> Homo sapiens
<400> 136
Leu Phe Ile Pro Gln Ile Thr Arg
1 5
<210> 137
<211> 7
<212> Белок
<213> Homo sapiens
<400> 137
Gly Pro Gly Glu Asp Phe Arg
1 5
<210> 138
<211> 9
<212> Белок
<213> Homo sapiens
<400> 138
Asn Tyr Gly Leu Leu Tyr Cys Phe Arg
1 5
<210> 139
<211> 8
<212> Белок
<213> Homo sapiens
<400> 139
Ser Val Glu Gly Ser Cys Gly Phe
1 5
<210> 140
<211> 11
<212> Белок
<213> Homo sapiens
<400> 140
Val Leu Thr His Ser Glu Leu Ala Pro Leu Arg
1 5 10
<210> 141
<211> 16
<212> Белок
<213> Homo sapiens
<400> 141
Leu Asn Trp Glu Ala Pro Pro Gly Ala Phe Asp Ser Phe Leu Leu Arg
1 5 10 15
<210> 142
<211> 15
<212> Белок
<213> Homo sapiens
<400> 142
Leu Ser Gln Leu Ser Val Thr Asp Val Thr Thr Ser Ser Leu Arg
1 5 10 15
<210> 143
<211> 8
<212> Белок
<213> Homo sapiens
<400> 143
Ala Val Leu His Ile Gly Glu Lys
1 5
<210> 144
<211> 20
<212> Белок
<213> Homo sapiens
<400> 144
Val Ser Trp Ser Leu Pro Leu Val Pro Gly Pro Leu Val Gly Asp Gly
1 5 10 15
Phe Leu Leu Arg
20
<210> 145
<211> 8
<212> Белок
<213> Homo sapiens
<400> 145
Tyr Leu Gly Glu Glu Tyr Val Lys
1 5
<210> 146
<211> 6
<212> Белок
<213> Homo sapiens
<400> 146
Val Glu Ile Asp Thr Lys
1 5
<210> 147
<211> 8
<212> Белок
<213> Homo sapiens
<400> 147
Glu Leu Pro Glu His Thr Val Lys
1 5
<210> 148
<211> 7
<212> Белок
<213> Homo sapiens
<400> 148
Val Leu Glu Pro Thr Leu Lys
1 5
<210> 149
<211> 10
<212> Белок
<213> Homo sapiens
<400> 149
Gly Gln Tyr Cys Tyr Glu Leu Asp Glu Lys
1 5 10
<210> 150
<211> 10
<212> Белок
<213> Homo sapiens
<400> 150
Val Asp Thr Val Asp Pro Pro Tyr Pro Arg
1 5 10
<210> 151
<211> 13
<212> Белок
<213> Homo sapiens
<400> 151
Phe Gly Phe Gly Gly Ser Thr Asp Ser Gly Pro Ile Arg
1 5 10
<210> 152
<211> 10
<212> Белок
<213> Homo sapiens
<400> 152
Leu Ile Glu Ile Ala Asn His Val Asp Lys
1 5 10
<210> 153
<211> 10
<212> Белок
<213> Homo sapiens
<400> 153
Leu Asn Ile Gly Tyr Ile Glu Asp Leu Lys
1 5 10
<210> 154
<211> 16
<212> Белок
<213> Homo sapiens
<400> 154
Ser Asn Pro Val Thr Leu Asn Val Leu Tyr Gly Pro Asp Leu Pro Arg
1 5 10 15
<210> 155
<211> 11
<212> Белок
<213> Homo sapiens
<400> 155
Ser Gly Val Asp Leu Ala Asp Ser Asn Gln Lys
1 5 10
<210> 156
<211> 10
<212> Белок
<213> Homo sapiens
<400> 156
His Ala Thr Leu Ser Leu Ser Ile Pro Arg
1 5 10
<210> 157
<211> 7
<212> Белок
<213> Homo sapiens
<400> 157
Asn Gly Val Asp Leu Asn Arg
1 5
<210> 158
<211> 258
<212> Белок
<213> Homo sapiens
<400> 158
Met Leu Pro Leu Cys Leu Val Ala Ala Leu Leu Leu Ala Ala Gly Pro
1 5 10 15
Gly Pro Ser Leu Gly Asp Glu Ala Ile His Cys Pro Pro Cys Ser Glu
20 25 30
Glu Lys Leu Ala Arg Cys Arg Pro Pro Val Gly Cys Glu Glu Leu Val
35 40 45
Arg Glu Pro Gly Cys Gly Cys Cys Ala Thr Cys Ala Leu Gly Leu Gly
50 55 60
Met Pro Cys Gly Val Tyr Thr Pro Arg Cys Gly Ser Gly Leu Arg Cys
65 70 75 80
Tyr Pro Pro Arg Gly Val Glu Lys Pro Leu His Thr Leu Met His Gly
85 90 95
Gln Gly Val Cys Met Glu Leu Ala Glu Ile Glu Ala Ile Gln Glu Ser
100 105 110
Leu Gln Pro Ser Asp Lys Asp Glu Gly Asp His Pro Asn Asn Ser Phe
115 120 125
Ser Pro Cys Ser Ala His Asp Arg Arg Cys Leu Gln Lys His Phe Ala
130 135 140
Lys Ile Arg Asp Arg Ser Thr Ser Gly Gly Lys Met Lys Val Asn Gly
145 150 155 160
Ala Pro Arg Glu Asp Ala Arg Pro Val Pro Gln Gly Ser Cys Gln Ser
165 170 175
Glu Leu His Arg Ala Leu Glu Arg Leu Ala Ala Ser Gln Ser Arg Thr
180 185 190
His Glu Asp Leu Tyr Ile Ile Pro Ile Pro Asn Cys Asp Arg Asn Gly
195 200 205
Asn Phe His Pro Lys Gln Cys His Pro Ala Leu Asp Gly Gln Arg Gly
210 215 220
Lys Cys Trp Cys Val Asp Arg Lys Thr Gly Val Lys Leu Pro Gly Gly
225 230 235 240
Leu Glu Pro Lys Gly Glu Leu Asp Cys His Gln Leu Ala Asp Ser Phe
245 250 255
Arg Glu
<210> 159
<211> 158
<212> Белок
<213> Homo sapiens
<400> 159
Met Glu Leu Ala Glu Ile Glu Ala Ile Gln Glu Ser Leu Gln Pro Ser
1 5 10 15
Asp Lys Asp Glu Gly Asp His Pro Asn Asn Ser Phe Ser Pro Cys Ser
20 25 30
Ala His Asp Arg Arg Cys Leu Gln Lys His Phe Ala Lys Ile Arg Asp
35 40 45
Arg Ser Thr Ser Gly Gly Lys Met Lys Val Asn Gly Ala Pro Arg Glu
50 55 60
Asp Ala Arg Pro Val Pro Gln Gly Ser Cys Gln Ser Glu Leu His Arg
65 70 75 80
Ala Leu Glu Arg Leu Ala Ala Ser Gln Ser Arg Thr His Glu Asp Leu
85 90 95
Tyr Ile Ile Pro Ile Pro Asn Cys Asp Arg Asn Gly Asn Phe His Pro
100 105 110
Lys Gln Cys His Pro Ala Leu Asp Gly Gln Arg Gly Lys Cys Trp Cys
115 120 125
Val Asp Arg Lys Thr Gly Val Lys Leu Pro Gly Gly Leu Glu Pro Lys
130 135 140
Gly Glu Leu Asp Cys His Gln Leu Ala Asp Ser Phe Arg Glu
145 150 155
<210> 160
<211> 54
<212> Белок
<213> Homo sapiens
<400> 160
Val Glu Val Lys Met Glu Gly Asp Ser Val Leu Leu Glu Val Asp Gly
1 5 10 15
Glu Glu Val Leu Arg Leu Arg Gln Val Ser Gly Pro Leu Thr Ser Lys
20 25 30
Arg His Pro Ile Met Arg Ile Ala Leu Gly Gly Leu Leu Phe Pro Ala
35 40 45
Ser Asn Leu Arg Leu Pro
50
<210> 161
<211> 36
<212> Белок
<213> Homo sapiens
<400> 161
Val Leu Arg Leu Arg Gln Val Ser Gly Pro Leu Thr Ser Lys Arg His
1 5 10 15
Pro Ile Met Arg Ile Ala Leu Gly Gly Leu Leu Phe Pro Ala Ser Asn
20 25 30
Leu Arg Leu Pro
35
<---