Изобретение относится к микробиологии, молекулярной биологии и может быть использовано в клинической микробиологии для расшифровки механизмов возникновения резистентности бактерий к антибиотикам.
Проблемы возникновения, распространения и эволюции резистентности микробов к антибиотикам находятся в центре внимания клинической микробиологии. Это связано с тем, что на фоне замедления разработки новых антимикробных препаратов наблюдается повсеместный рост резистентности к ним и появление панрезистентных штаммов микробов. С начала 1990-х гг. наблюдается значительная тенденция к росту числа инфекций, связанных с грамотрицательными возбудителями, которые обладают фенотипом множественной лекарственной устойчивости. Грамотрицательные патогены, такие как Klebsiella pneumoniae, A. baumannii, P. aeruginosa, обладают свойством приобретать устойчивость к антимикробным препаратам. Развитие и совершенствование молекулярно-генетических технологий внесло существенный вклад в формирование базы знаний, касающихся механизмов устойчивости к антибиотикам, их эволюции и распространения. Актуальные данные о частоте возникновения устойчивости, молекулярных механизмах возникновения резистентности конкретного вида возбудителя инфекционного процесса, лежат в основе эффективности антибактериальной терапии. Описаны различные механизмы, опосредующие устойчивость к антибиотикам: модификация мишени действия, инактивация антибиотика, активное выведение антибиотика из микробной клетки - эффлюкс, нарушение проницаемости внешних структур микробной клетки, формирование метаболического "шунта". Большую роль в распространении генетических детерминант антибиотикорезистентности, повреждении генов, кодирующих структуры бактериальной клетки, играют мобильные генетические элементы. Известен фрагмент ДНК, обнаруженный у Pseudomonas aeruginosa, содержащий нуклеотидную последовательность ISPa195, инактивирующий ген порина OprD, что приводит к резистентности к карбапенемным антибиотикам (Y Bocharova, 2019, doi: 10.1016/j.jgar.2019.01.016).
Известен фрагмент ДНК, обнаруженный у Acinetobacter baumannii, содержащий нуклеотидную последовательность ISAba10, внедренный в ген blaOXA-23, что приводит к резистентности к карбапенемным антибиотикам (Y Lee, 2011, doi: 10.1128/ААС.01672-09).
Известен фрагмент ДНК, обнаруженный у A. baumannii, содержащий нуклеотидную последовательность ISAba11, внедренный в гены lpxA и lpxC, что приводит к резистентности к колистину, поскольку у бактерий наблюдается полная утрата липополисахарида клеточной стенки (JH Moffatt, 2011, doi: 10.1128/ААС.01732-10).
Известны три фрагмента ДНК, обнаруженные у Burkholderia glumae, содержащие нуклеотидные последовательности IS1417, IS1418, и IS1419, способные активировать экспрессию неогена вектора-ловушки pSHI1063 (A Hasebe, 2000, doi: 10.1128/ААС.01672-09).
Известен рекомбинантный полипептид с активностью цефалоспоринацетилгид-ролазы, имеющий аминокислотную последовательность общей формулы [HY]n, где n 4 или 8; Y аминокислотная последовательность с мол.м. примерно 35 кД, кодируемый геном цефалоспоринацетилгидролазы, происходящим от Bacillus subtilis SHS 0133 (Ferm BP-2755. При получении полипептида, обладающего активностью цефалоспорингидролазы, ведут культивирование клеток Escherichia coli, трансформированных рекомбинантной молекулой ДНК, содержащей ДНК-фрагмент, полученный из генома В. subtilis SHS 0133, продуцирующий указанный полипептид (Патент РФ №2103364).
Известен Фрагмент ДНК, который кодирует промотор, обеспечивающий регулируемую кальцием экспрессию полипептидов, в частности ферментов в клетках Streptomyces (Патент РФ №2112802).
Известна рекомбинантная плазмидная ДНК pMind-vapC, представляющую собой плазмиду pMind, в которую клонирована последовательность. Рекомбинантная плазмидная ДНК pMind-vapC позволяет осуществлять гиперэкспрессию токсина VapC в штаммах Mycobacterium smegmatis и Mycobacterium tuberculosis (Патент РФ №2524143), выбранный нами в качестве прототипа.
Задачей изобретения является фрагмент ДНК, содержащий нуклеотидную последовательность с инвертированными повторами MITEKpnl у колистин-резистентных штаммов К. pneumoniae, которая может быть использована в качестве референсной при поиске мутаций в генах бактерий и расшифровать механизмы возникновения резистентности бактерий к антибиотикам.
Технический результат, на достижение которого направлена данная нуклеотидная последовательность MYYEKpnl, заключается в ее способности к интеграции и перемещениям внутри генома, а так же способности изменять структуру, функцию и экспрессию генов, в результате чего ее обнаружение в бактериальном геноме поможет расшифровать механизмы возникновения резистентности бактерий к антибиотикам.
Сущность изобретения заключается в создании нуклеотидной последовательности с инвертированными повторами MYYEKpnl у колистин-резистентных штаммов K. pneumoniae, которая может быть использована в качестве референсной при поиске мутаций в генах бактерий и расшифровать механизмы возникновения резистентности бактерий к антибиотикам. С помощью секвенирования была получена нуклеотидная последовательность MITEKpnl. Для выделения ДНК использовали суточную культуру штамма K. pneumoniae, резистентного к колистину, полученную при посеве на агар Uri-select (BioRad, США). Экстракцию ДНК из бактериальных культур выполняли с помощью коммерческого набора реагентов DNeasy Blood & Tissue Kit (Qiagen, Германия) в соответствии с инструкцией производителя. Для выявления и секвенирования mgrB гена использовались праймеры, предложение 62. Ген mgrB выявляли с помощью полимеразной-цепной реакции (ПЦР). Результаты ПЦР реакции оценивали путем проведения горизонтального электрофореза в 2% агарозном геле. Подготовка матриц для секвенирования включала амплификацию гена mgrB. Очистку ПЦР-продуктов от избытка праймеров, димеров праймеров и дНТФ проводили ферментативным разрушением с помощью экзонуклеазы I и щелочной фосфатазы (SAP, Affymetrix, США) при температуре 37°С в течение 30 мин. с последующей инактивацией при 85°С. Секвенирование проводили методом Сенгера с использованием наборов реагентов «BigDye™ Terminator v3.1 Cycle Sequencing Kit» (Thermo Fisher Scientific) и оборудования фирмы Applied Biosystems (США) по методике, описанной производителем. Полученные в результате секвенирования нуклеотидные последовательности гена mgrB обрабатывали с помощью программы SeqMan.
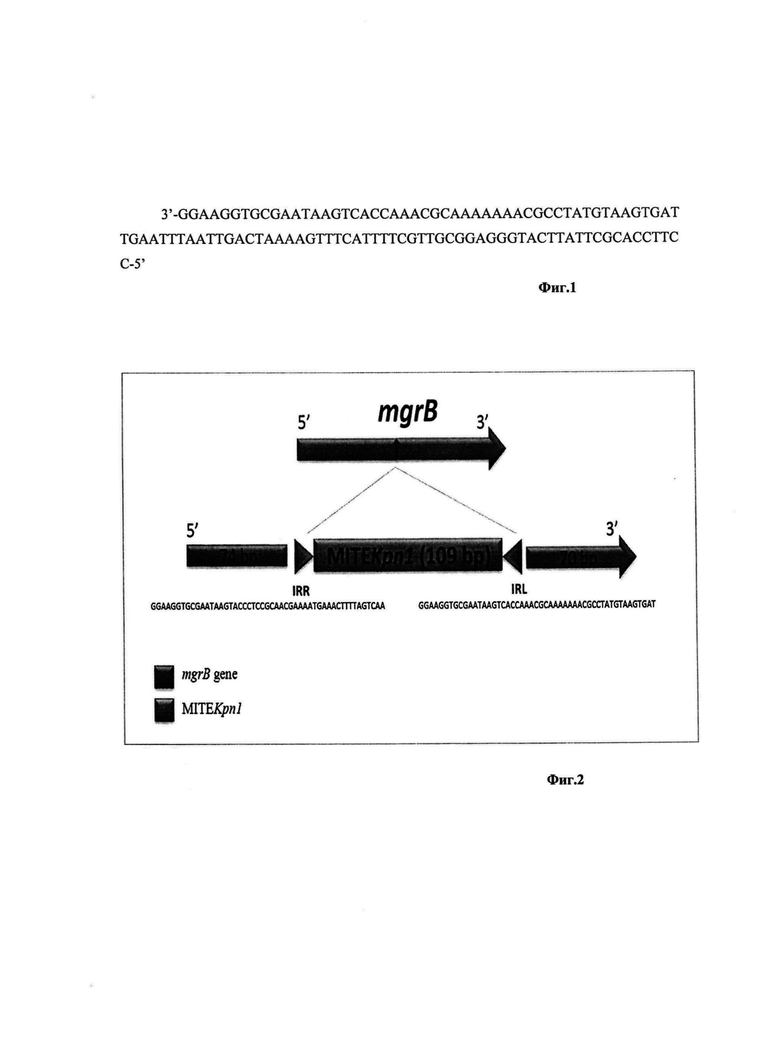
Нуклеотидная последовательность MITEKpnl приведена на Фиг. 1.
Нуклеотидная последовательность нового вставочного элемента MITEKpnl, встроенного в mgrB ген, была внесена в международную базу данных GenBank под номером МK241841 (https://www.ncbi.nlm.nih.gov/nuccore/MK241841.2/). Нуклеотидная последовательность MITEKpnl внесена в международную базу данных «IS-finder» под номером СР018364 (https://www-is.biotoul.fr/scripts/ficheIS.php?name=MITEKpnl).
Внесенная последовательность стала референсной для поиска аналогичных вставочных элементов и выявления нарушений структуры генов. Рекомбинантная плазмидная ДНК pMind - vapC, обеспечивающая гиперэкспрессию гена vapC в клетках М. tuberculosis, представляющая собой плазмиду pMind, в которую клонирована нуклеотидная последовательность. Установлена и описана нуклеотидная последовательность фрагмента ДНК, нарушающая структуру и функцию гена mgrB у колистин-резистентного штамма K. pneumoniae. Данный фрагмент ДНК относится к группе миниатюрных мобильных элементов с инвертированными повторами (Miniature Inverted Repeat Transposable Element, MITE), которому было присвоено название MITEKpnl. MITE представляют собой последовательности ДНК, способные к интеграции и перемещениям внутри хозяйского генома, они играют важную роль в изменении структуры генома, функций и экспрессии генов. MITE обычно не имеют открытых рамок считывания, поэтому переносятся через транспозазы, закодированные автономными ДНК-транспозонами. Из-за того, что MITE имеет малый размер их передвижение в геноме до конца не изучено и имеются трудности в их идентификации и исследовании. Обнаруженный МIТЕKpnI состоит из 109 пар нуклеотидов (пн) принадлежит к семейству вставочных элементов IS5-family, не имеет открытых рамок считывания, содержит прямые повторы (direct repeat, DR) размером 4 пн (СТАА). Во время исследования, фрагмент ДНК, описанный как MITEKpnl, был обнаружен в гене mgrB, который кодирует маленький трансмембранный белок MgrB, негативный регулятор системы PhoPQ (Фиг. 2). При фосфорилировании регуляторного белка PhoP происходит транскрипция mgrB. Белок MgrB подавляет экспрессию гена, кодирующего PhoQ. Таким образом, происходит регуляция двухкомпонентной системы PhoPQ по типу отрицательной обратной связи. При инактивации mgrB происходит гиперактивация системы PhoPQ, что ведет к активации транскрипции pmrHFIJKLM оперона и присоединению L-Ara4N к липополисахариду клеточной стенки граммотрицательных бактерий, что в конечном итоге приводит к развитию резистентности к антибиотикам группы полимиксинов (колистин). Группа полимиксинов, а именно колистин, вызывает значительный интерес в лечении заболеваний, вызванных грамотрицательными бактериями с множественной лекарственной устойчивостью, поскольку сохраняет активность в отношении данных возбудителей. Однако, в последнее время наблюдается быстрое распространение резистентности и к данному препарату. На сегодняшний день в литературе описаны молекулярные механизмы, лежащие в основе появления устойчивости к колистину, но обнаруженный MYTEKpn1, который являлся причиной резистентности к колистину у исследованных штаммов, описан впервые. Поскольку такие вставочные элементы могут мигрировать в геноме, может встречаться сразу несколько их копий в разных генах, а также, поскольку вставочные элементы распространяются посредством горизонтального переноса между бактериями разных видов, понятно, что для поиска таких модификаций необходима референсная последовательность.
Использование описанной нуклеотидной последовательности, способной к интеграции и перемещениям внутри генома, а так же способной изменять структуру, функцию и экспрессию гена, ее обнаружение в бактериальном геноме поможет расшифровать механизмы возникновения резистентности бактерий к антибиотикам, что будет способствовать рациональному выбору антимикробных препаратов и разработке мер по преодолению устойчивости. Изобретение может быть использовано при анализе данных полногеномного секвенирования.
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ идентификации генов клинически значимых семейств β-лактамаз у грамотрицательных микроорганизмов | 2015 |
|
RU2608651C1 |
| РЕКОМБИНАНТНАЯ ПЛАЗМИДНАЯ ДНК pMind-vapC, СОДЕРЖАЩАЯ НУКЛЕОТИДНУЮ ПОСЛЕДОВАТЕЛЬНОСТЬ, КОДИРУЮЩУЮ ГЕН vapC MSMEG_1284 | 2012 |
|
RU2524143C2 |
| Синтетические олигонуклеотидные праймеры для идентификации штаммов и изолятов бактерии Acinetobacter и определения устойчивости к бета-лактамным антибиотикам и способ их применения | 2016 |
|
RU2646107C1 |
| Штамм метициллинрезистентного Staphylococcus aureus (MRSA), характеризующийся устойчивостью к цефтаролину и используемый в качестве контрольной тест-культуры для определения чувствительности к бета-лактамным антибиотикам (варианты) | 2022 |
|
RU2798788C1 |
| ПОЛИРЕЗИСТЕНТНЫЙ ШТАММ БАКТЕРИЙ ESCHERICHIA COLI ДЛЯ ОПРЕДЕЛЕНИЯ БАКТЕРИЦИДНОГО ДЕЙСТВИЯ АНТИБАКТЕРИАЛЬНЫХ ПРЕПАРАТОВ В ВЕТЕРИНАРИИ | 2022 |
|
RU2797383C1 |
| ШТАММ БАКТЕРИЙ SALMONELLA ENTERICA (ВКШМ-Б-849М) В КАЧЕСТВЕ КОНТРОЛЬНОГО ШТАММА ДЛЯ МИКРОБИОЛОГИЧЕСКИХ И МОЛЕКУЛЯРНО-ГЕНЕТИЧЕСКИХ ИССЛЕДОВАНИЙ В ОПРЕДЕЛЕНИИ АНТИБИОТИКОРЕЗИСТЕНТНОСТИ | 2020 |
|
RU2759396C1 |
| Олигонуклеотиды для определения 15BC серотипа Streptococcus pneumoniae | 2021 |
|
RU2795316C1 |
| Олигонуклеотиды для определения 12FAB серотипа Streptococcus pneumoniae | 2021 |
|
RU2795021C1 |
| Олигонуклеотиды для определения 22FA серотипа Streptococcus pneumoniae | 2021 |
|
RU2802203C2 |
| Штамм бактерий Klebsiella pneumoniae, используемый в качестве тест-культуры при детекции гена NDM методом полимеразной цепной реакции для точной диагностики и назначения антибактериальной терапии клебсиелёзных инфекций | 2022 |
|
RU2797025C1 |
Изобретение относится к области биотехнологии. Путем секвенирования была получена нуклеотидная последовательность МITЕKpnl. Для выделения ДНК использовали суточную культуру штамма К. pneumoniae, резистентного к колистину, полученную при посеве на агар Uri-select (BioRad, США). Экстракцию ДНК из бактериальных культур выполняли с помощью коммерческого набора реагентов DNeasy Blood & Tissue Kit (Qiagen, Германия) в соответствии с инструкцией производителя. Установлена и описана нуклеотидная последовательность фрагмента ДНК, нарушающая структуру и функцию гена mgrB у колистин-резистентного штамма K. pneumoniae. Использование описанной нуклеотидной последовательности MYTEKpnl:3GGAAGGTGCGAATAAGTCАССAAACGCААА АAAACGCCTATGTAAGTGATTGAАTTTAATT GACTAAAAGTTTCATTTTCGTTGCGG AGGGTACTTATTCGCАССТТСС-5', способной к интеграции и перемещениям внутри генома, а также способной изменять структуру, функцию и экспрессию гена, ее обнаружение в бактериальном геноме поможет расшифровать механизмы возникновения резистентности бактерий к антибиотикам, что будет способствовать рациональному выбору антимикробных препаратов и разработке мер по преодолению устойчивости. 2 ил.
Фрагмент ДНК, содержащий нуклеотидную последовательность 3'-GGAAGGTGCGAATAAGTCАССAAACGCAAAAAAACGCCTATGTAAGTGATTGAATTTAA TTGACTAAAAGTTTCATTTTCGTTGCGGAGGGTACTTATTCGCACCTTCC-5' с инвертированными повторами MITEKpnl у колистин-резистентных штаммов Klebsiella pneumoniae, которая может быть использована в качестве референсной при поиске мутаций в генах бактерий и расшифровать механизмы возникновения резистентности бактерий к антибиотикам.
| РЕКОМБИНАНТНАЯ ПЛАЗМИДНАЯ ДНК pMind-vapC, СОДЕРЖАЩАЯ НУКЛЕОТИДНУЮ ПОСЛЕДОВАТЕЛЬНОСТЬ, КОДИРУЮЩУЮ ГЕН vapC MSMEG_1284 | 2012 |
|
RU2524143C2 |
| YULIYA BOCHAROVA и др | |||
| Регулятор давления для автоматических тормозов с сжатым воздухом | 1921 |
|
SU195A1 |
| Станок для придания концам круглых радиаторных трубок шестигранного сечения | 1924 |
|
SU2019A1 |
| Станок для придания концам круглых радиаторных трубок шестигранного сечения | 1924 |
|
SU2019A1 |
Авторы
Даты
2020-11-11—Публикация
2020-03-16—Подача