Изобретение относится к области биотехнологии, в частности к определению 15 ВС серотипа Streptococcus pneumoniae с использованием полимеразной цепной реакции в режиме реального времени (ПЦР-РРВ) и может использоваться для идентификации возбудителей пневмококковых инфекций (ПИ) и оценки эффективности проводимых иммунопрофилактических мероприятий.
S. pneumoniae является грамположительным анаэробным видом бактерий. В зависимости от химических свойств полисахаридной капсулы выделяют около 50 серогрупп, образующих не менее 100 серотипов, многие из которых ассоциированы с инвазивными ПИ. Важным элементом эпидемиологического надзора за пневмококковыми инфекциями является микробиологический мониторинг, включающий антигенную и генетическую характеристику возбудителей ПИ, а также данные о чувствительности к антибиотикам. Антигенная характеристика возбудителей заключается в определении серотипов, что позволяет оценить эффективность существующих поливалентных вакцин. В России широкое применение получили 13-валентная конъюгированная пневмококковая вакцина (PCV13, «Превенар 13») и 23-валентная полисахаридная вакцина (PPV23, «Пневмовакс 23»). Определение спектра серотипов возбудителей ПИ позволяет планировать иммунопрофилактические мероприятия и оценивать их эффективность в группах лиц, вовлеченных в эпидемический процесс.
Для определения серотипов S. pneumoniae используются серологические способы, основанные на реакции набухания полисахаридной капсулы или реакции латекс-агглютинации. Широко используются и основанные на ПЦР способы детекции серотип-специфических последовательностей cps-локуса, такие как гены полисахаридной полимеразы wzy и флиппазы wzx, а также другие гены, участвующие в биосинтезе капсульного полисахарида (wcwV, galU, wciP, wzg) [Bentley, S.D. Genetic analysis of the capsular biosynthetic locus from all 90 pneumococcal serotypes / S.D. Bentley, D.M. Aanensen, A. Mavroidi et al. // PLoS Gene. - 2006. - Vol.2. - №3. - P.262-269. doi: 10.1371/journal.pgen.0020031; Selva, L. Rapid and easy identification of capsular serotypes of Streptococcus pneumoniae by use of fragment analysis by automated fluorescence-based capillary electrophoresis / L. Selva, E. del Amo, P. Brotons et al. // J. Clin. Microbiol. - 2012. -Vol.50. №11. P.3451-3457. doi: 10.1128/JCM.01368-12].
В 2006 году R. Pai и соавторы предложили алгоритм серотипирования S. pneumoniae, основанный на последовательных мультиплексных ПЦР [Pai, R. Sequential multiplex PCR approach for determining capsular serotypes of Streptococcus pneumoniae isolates / R. Pai, R. E. Gertz, B. Beall // J. Clin. Microbiol. - 2006. - Vol.44. - №1. - P.124-131. doi:10.1128/JCM.44.1.124-131.2006]. К недостаткам данного подхода относится необходимость использования электрофоретической детекции продуктов ПЦР в агарозном геле, что, наряду с другими недостатками включения данного этапа, может осложнять дифференциацию серотипов при проведении мультиплексной ПЦР.
Из уровня техники также известен способ определения 29 серотипов [Yun, К. W. Streptococcus pneumoniae type determination by multiplex polymerase chain reaction / K. W. Yun, E. Y. Cho, К. B. Hong et al. // Journal of Korean medical science. - 2011. - Vol.26. - №8. - P.971-978. doi:10.3346/jkms.2011.26.8.971]. Недостатком данного способа также является этап электрофореза.
Оптимальным способом одновременного определения нескольких серотип-специфических мишеней является ПЦР-РРВ с использованием флуоресцентно меченых зондов. Несмотря на преимущества ПЦР-РРВ для определения серотипов, эффективность ее применения не всегда очевидна, что обусловлено постоянной адаптацией возбудителей под давлением популяционного иммунитета, в том числе обусловленного иммунопрофилактикой с использованием поливалентных вакцин.
Центрами по контролю и профилактике заболеваний США (Centers for Disease Control and Prevention) рекомендованы последовательности олигонуклеотидов для определения серотипов S. pneumoniae с использованием ПЦР-РРВ и опубликованы алгоритмы проведения серотипирования для Латинской Америки, Африки, Азии и США [Real-time PCR Deduction of Pneumococcal Serotypes or Serogroups PCR based methods for identification of S. pneumoniae serotypes/serogroups. URL: www.cdc.gov/streplab/pneumococcus/resources.html? CDC_AA_ refVal=https%3A%2F%2Fwww.cdc.gov%2Fstreplab%2Fpcr.html (дата обращения: 01.09.2021)]. Рекомендованные протоколы постановки ПЦР-РРВ предполагают определение трех серотип-специфических мишеней в одной реакции. Различия в способах серотипирования связаны с тем, что частота обнаружения тех или иных серотипов варьирует в зависимости от региона и параметров эпидемического процесса на каждой территории (доля инвазивных и неинвазивных форм ПИ, спектр циркулирующих серотипов и их изменение в зависимости от проводимых иммунопрофилактических мероприятий), которые определяют особенности проведения микробиологического мониторинга S. pneumoniae.
Детекция дополнительных серотип-специфических мишеней, также как и исключение из анализа мишеней для серотипов, не характерных для наблюдаемой территории, позволяет повысить долю определяемых серотипов и эффективность лабораторного исследования в целом. Уменьшение доли штаммов, нетипируемых с помощью стандартных серологических или основанных на ПЦР методик, способствует накоплению информации об эпидемиологических особенностях циркулирующих возбудителей для планирования и контроля эффективности программ по вакцинации. В связи с этим существует потребность в разработке олигонуклеотидов для выявления 15 ВС серотипа S. pneumoniae: праймеров и зонда для проведения ПЦР-РРВ в мультиплексном формате.
Технический результат заявляемого изобретения направлен на выявление ДНК 15 ВС серотипа S. pneumoniae с высокой степенью специфичности в образцах биологического материала посредством таких олигонуклеотидов праймеров и флоуресцентного зонда, которые позволяют эффективно, определять серотип-специфическую последовательность ДНК с использованием широко распространенных методик и доступных материалов.
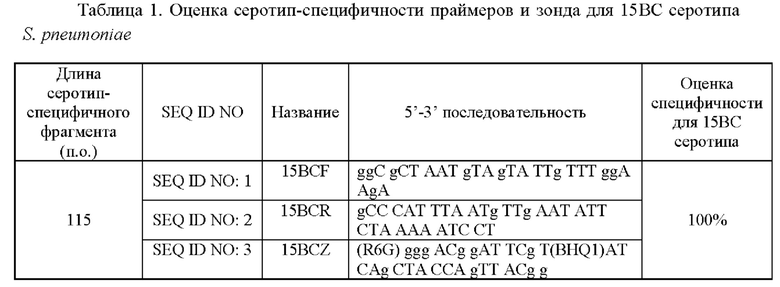
Технический результат достигается за счет применения в ПЦР-РРВ синтезированных олигонуклеотидов для определения 15 ВС серотипа S. pneumoniae, имеющих следующий олигонуклеотидный состав:
прямой праймер 15BCF SEQ ID NO: 1;
обратный праймер 15BCR SEQ ID NO: 2;
флоуресцентный зонд 15BCZ - SEQ ID NO: 3.
Праймеры представляют собой последовательности олигонуклеотидов для амплификации серотип-специфического фрагмента ДНК, флоуресцентный зонд является олигонуклеотидом, содержащим флуорофор и гаситель флуоресценции, позволяющим детектировать амплифицированный фрагмент.
Заявляемые олигонуклеотиды разработаны на основе генов S. pneumoniae, входящих в cps-локус [Bentley, S.D. Genetic analysis of the capsular biosynthetic locus from all 90 pneumococcal serotypes / S.D. Bentley, D.M. Aanensen, A. Mavroidi et al. // PLoS Gene. 2006. Vol.2. №3. P.262-269. doi: 10.1371/journal.pgen.0020031]. С этой целью были проанализированы фрагменты последовательностей генома S. pneumoniae (cps-локуса) 15 ВС серотипа для определения консервативных участков ДНК, не имеющих гомологии с фрагментами cps-локуса представителей других серотипов S. pneumoniae. В результате проведенного анализа был выбран гомологичный для 15 ВС серотипа S. pneumoniae фрагмент cps-локуса, к которому были подобраны праймеры и зонд для амплификации фрагмента 115 пар оснований: прямой 15BCF - SEQ ID NO: 1; обратный 15BCR- SEQ ID NO: 2 и флоуресцентный зонд 15BCZ - SEQ ID NO: 3.
Заявляемое изобретение является результатом работы в рамках исследования российских штаммов S. pneumoniae, проделанной в ФБУН ЦНИИ Эпидемиологии Роспотребнадзора (Москва, Россия).
Для подбора последовательностей-мишеней - мест посадки олигонуклеотидов были использованы фрагменты референсных геномов 15 ВС серотипа S. pneumoniae, взятые из базы данных GenBank. Для поиска консервативных последовательностей были использованы современные алгоритмы in silico анализа нуклеотидных последовательностей и программы, находящиеся в открытом доступе, включая Mega и Unipro UGENE, олигокалькуляторы (Oligo Calculators) Thermo Fisher Scientific. Составляют перечень консервативных участков, характерных только для 15 ВС серотипа S. pneumoniae, к которым подбирают олигонуклеотидные последовательности прямого и обратного праймеров, а также флоуресцентный зонд. Упомянутые олигонуклеотидные последовательности приведены в Таблице 1.
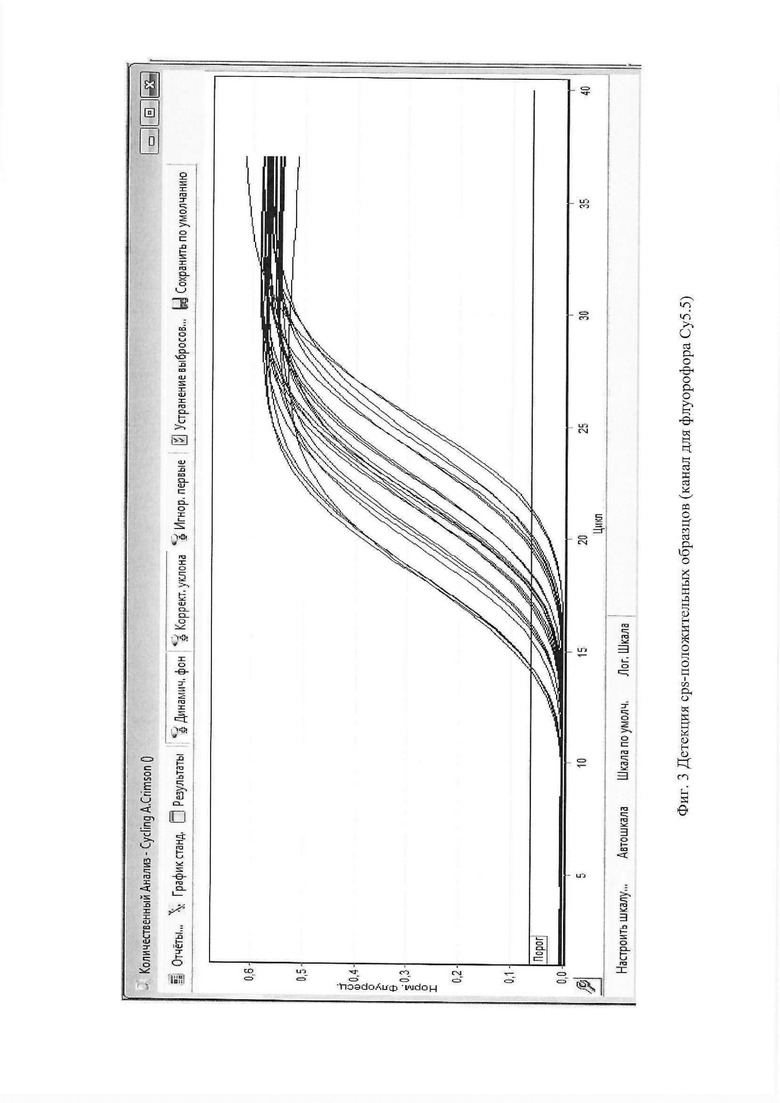
В качестве положительного внутреннего контроля используется фрагмент срт-локуса общего для всех серотипов SEQ ID NO: 4-6, с праймерами cpsA-f и cpsA-r [Pai, R. Sequential multiplex PCR approach for determining capsular serotypes of Streptococcus pneumoniae isolates / R. Pai, R.E. Gertz, B. Beall // J. Clin. Microbiol. 2006. Vol.44. №1. P.124-131. doi: 10.1128/JCM.44.1.124-131.2006] и зондом cpsA-Z2 [Миронов, К.О. Методика ПЦР в режиме реального времени для определения серотипов Streptococcus pneumoniae / К.О. Миронов, А.Е. Платонов, Е.А. Дунаева и др. // Журнал микробиологии, эпидемиологии и иммунобиологии. 2014. №1. С.41-48.]. Для детекции cps-положительных образцов используют канал для флуорофора Су5.5.
Анализ заявляемых последовательностей с использованием ресурса Primer BLAST продемонстрировал, что прямой 15BCF (SEQ ID NO: 1) и обратный 15BCR (SEQ ID NO: 2) праймеры амплифицируют консервативный для 15 ВС серотипа участок со 100% специфичностью.
где R6G - флуорофор, BHQ1 - гаситель флуоресценции.
Заявляемое решение также поясняется:
на Фиг 1, на которой представлен консервативный участок генома 15 ВС серотипа S. pneumoniae и места посадки праймеров и флоуресцентного зонда, где:
1 - прямой праймер 15BCF (SEQ ID NO: 1),
2 - флоуресцентный зонд 15BCZ, (SEQ ID NO: 3),
3 - обратный праймер 15BCR, (SEQ ID NO: 2);
на Фиг. 2, где показана детекция серотипа 15 ВС (канал для флуорофора R6G);
на Фиг. 3, где показана детекция cps-положительных образцов (канал для флуорофора Су5.5).
В качестве биологического материала предпочтительно использовать спинномозговую жидкость или образцы культур S. pneumoniae.
Выделение ДНК из биологического материала или штаммов проводят в соответствии с МУ 1.3.2569-09 «Организация работ лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Выделение ДНК из биологического материала производят с помощью комплекта реагентов в соответствии с инструкцией производителя. Для выделения ДНК может быть использован комплект реагентов «РИБО-преп» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) или любой аналогичный набор для выделения ДНК. Оптимальная концентрация ДНК - 103-105 копий в 10 мкл.
ПЦР-РРВ проводится с применением заявляемых олигонуклеотидов - праймеров и зонда, для серотип-специфичной мишени 15 ВС серотипа, представленных в Таблице 1.
ПЦР-РРВ проводят при следующих условиях:
Общий объем реакционной смеси - 25 мкл.
Компоненты ПЦР смешиваются следующим образом:
(a) 10 мкл смеси, содержащей:
- олигонуклеотидные праймеры SEQ ID NO: 1, 2, 4, 5 - по 0,5 мкМ;
- флоуресцентный зонд SEQ ID NO: 3,6- 0,2 мкМ;
- dNTPs - 0,44 мМ.
(b) реактив, содержащий рекомбинантный фермент Taq ДНК-полимеразы, например 0,5 мкл реактива «Полимераза TaqF» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) или любого аналогичного коммерческого набора в соответствии с инструкцией производителя.
(c) ПЦР-буфер, содержащий MgCl2, например 4,5 мкл ПЦР-буфера «ОТ-ПЦР-смесь-2 FEP/FRT» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) или любого аналогичного коммерческого набора в соответствии с инструкцией производителя.
(d) выделенная ДНК - 10 мкл.
ПЦР-РРВ проводят с флуоресцентной детекцией на приборе «Rotor-Gene Q» («Qiagen», Германия) или на любом другом приборе с 5 каналами детекции.
Амплификацию проводят по следующей программе: 1 цикл 95°С в течение 15 минут, 5 циклов при температуре 95°С - 5 секунд / 60°С - 20 секунд / 72°С - 5 секунд, затем 40 циклов при температуре 95°С - 5 секунд / 60°С - 20 сек / 72°С - 5 секунд. Детекция флуоресценции проводится на этапе 60°С по каналу для флуорофора R6G.
Для анализа результатов задают пороговую линию, соответствующую величине 8-10% от максимального сигнала положительного образца. Образцы, для которых кривые флуоресценции пересекают пороговую линию, и при этом кинетика накопления флуоресцентного сигнала является экспоненциальной, являются положительными, то есть содержат серотип-специфическую мишень для 15 ВС серотипа S. pneumoniae.
Реализация заявляемого изобретения поясняется следующими примерами:
Пример 1. Получение олигонуклеотидов для определения 15 ВС серотипа S. pneumoniae
Для подбора последовательностей-мишеней - мест посадки олигонуклеотидов были использованы фрагменты референсных геномов 15 ВС серотипа S. pneumoniae, взятые из базы данных GenBank. Для поиска консервативных последовательностей были использованы современные алгоритмы in silico анализа нуклеотидных последовательностей и программы, находящиеся в открытом доступе, включая Mega и Unipro UGENE, олигокалькуляторы (Oligo Calculators) Thermo Fisher Scientific. Был составлен перечень консервативных участков, характерных только для 15 ВС серотипа S. pneumoniae, к которым были подобраны олигонуклеотидные последовательности прямого 15BCF и обратного 15BCR праймеров, а также флоуресцентный зонд 15BCZ.
В качестве положительного внутреннего контроля используется фрагмент cps-локуса общего для всех серотипов S. pneumoniae SEQ ID NO: 4-6.
Анализ упомянутых последовательностей с использованием ресурса Primer BLAST показал, что прямой 15BCF и обратный 15BCR праймеры амлифицируют консервативный для 15 ВС серотипа участок со 100% специфичностью.
Олигонуклеотиды для определения 15 ВС серотипа S. pneumoniae - прямой праймер 15BCF, обратный праймер 15BCR, флоуресцентный зонд 15BCZ, представлены уникальными последовательностями SEQ ID NO: 1-3 соответственно.
Пример 2. Детекция 15 ВС серотипа S. pneumoniae методом ПЦР-РРВ
Определение 15 ВС серотипа S. pneumoniae проводят методом ПЦР-РРВ при следующих условиях:
Общий объем реакционной смеси - 25 мкл.
Компоненты ПЦР смешиваются следующим образом:
(a) 10 мкл смеси, содержащей:
- олигонуклеотидные праймеры SEQ ID NO: 1, 2, 4, 5 по 0,5 мкМ;
- флоуресцентный зонд SEQ ID NO: 3,6- 0,2 мкМ;
- dNTPs - 0,44 мМ.
(b) 0,5 мкл реактива «Полимераза TaqF» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия).
(c) 4,5 мкл ПЦР-буфера «ОТ-ПЦР-смесь-2 FEP/FRT» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия).
(d) выделенная ДНК - 10 мкл.
ПЦР-РРВ проводили с флуоресцентной детекцией на приборе с 5 каналами детекции - «Rotor-Gene Q» («Qiagen», Германия).
Подготовленный описанным способом материал, содержащий уникальные олигонуклеотидные последовательности SEQ ID NO: 1-3 используют для определения 15 ВС серотипа S. pneumoniae в биологических образцах.
Пример 3. Обнаружение 15 ВС серотипа S. pneumoniae в образцах биологического материала
Для определения 15 ВС серотипа S. pneumoniae было выбрано 45 смывов бактериальных культур. Выделение ДНК из биологического материала проводили в соответствии с МУ 1.3.2569-09 «Организация работ лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для выделения ДНК использовался набор реагентов «РИБО-преп» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия), концентрация ДНК - 103 копий в 10 мкл.
ПЦР-РРВ проводили в условиях, описанных в Примере 2, с использованием уникальных олигонуклеотидных последовательностей SEQ ID NO: 1-3.
Амплификацию проводили по следующей программе: 1 цикл 95°С в течение 15 минут, 5 циклов при температуре 95°С - 5 секунд / 60°С - 20 секунд / 72°С - 5 секунд, затем 40 циклов при температуре 95°С - 5 секунд / 60°С - 20 сек / 72°С - 5 секунд. Детекцию флуоресценции проводили на этапе 60°С по каналу для флуорофора R6G. Для анализа результатов задали пороговую линию, соответствующую величине 10% от максимального сигнала положительного образца. Для восьми образцов кривые флуоресценции пересекли пороговую линию и, при этом кинетика накопления флуоресцентного сигнала была экспоненциальной, что свидетельствует о том, что данные образцы содержат серотип-специфическую мишень 15 ВС серотипа S. pneumoniae.
Таким образом, из всей выборки было выявлено восемь образцов, принадлежащих к 15 ВС серотипу S. pneumoniae. Принадлежность данных образцов к 15 ВС серотипу S. pneumoniae подтверждена анализом данных полногеномного секвенирования с использованием программ «SeroBA» и «PneumoCaT».
Пример 4. Обнаружение 15 ВС серотипа S. pneumoniae в образцах биологического материала
Для определения 15 ВС серотипа S. pneumoniae было выбрано 38 смывов с бактериальных культур. Выделение ДНК из биологического материала проводили в соответствии с МУ 1.3.2569-09 «Организация работ лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для выделения ДНК использовался набор реагентов «РИБО-преп» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия), концентрация ДНК 105 копий в 10 мкл.
ПЦР-РРВ проводили в условиях, описанных в Примере 2, с использованием уникальных олигонуклеотидных последовательностей SEQ ID NO: 1-3.
Амплификацию проводили по следующей программе: 1 цикл 95°С в течение 15 минут, 5 циклов при температуре 95°С - 5 секунд / 60°С - 20 секунд / 72°С - 5 секунд, затем 40 циклов при температуре 95°С - 5 секунд / 60°С - 20 сек / 72°С - 5 секунд. Детекцию флуоресценции проводили на этапе 60°С по каналу для флуорофора R6G. Для анализа результатов задали пороговую линию, соответствующую величине 9% от максимального сигнала положительного образца. Для трех образцов кривые флуоресценции пересекли пороговую линию и при этом кинетика накопления флуоресцентного сигнала была экспоненциальной, что свидетельствует о том, что данные образцы содержат серотип-специфическую мишень для 15 ВС серотипа S. pneumoniae.
Таким образом, из всей выборки было выявлено три образца, принадлежащих к 15 ВС серотипу S. pneumoniae. Принадлежность данных образцов к 15 ВС серотипу S. pneumoniae подтверждена анализом данных полногеномного секвенирования с использованием программ «SeroBA» и «PneumoCaT».
Пример 5. Обнаружение 15 ВС серотипа S. pneumoniae в образцах биологического материала
Для определения 15 ВС серотипа S. pneumoniae было выбрано 50 смывов бактериальных культур. Выделение ДНК из биологического материала проводили в соответствии с МУ 1.3.2569-09 «Организация работ лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для выделения ДНК использовался набор реагентов «РИБО-преп» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия), концентрация ДНК 103 копий в 10 мкл.
ПЦР-РРВ проводили в условиях, описанных в Примере 2, с использованием уникальных олигонуклеотидных последовательностей SEQ ID NO: 1-3.
Амплификацию проводили по следующей программе: 1 цикл 95°С в течение 15 минут, 5 циклов при температуре 95°С - 5 секунд / 60°С - 20 секунд / 72°С - 5 секунд, затем 40 циклов при температуре 95°С - 5 секунд / 60°С - 20 сек / 72°С - 5 секунд. Детекцию флуоресценции проводили на этапе 60°С по каналу для флуорофора R6G. Для анализа результатов задали пороговую линию, соответствующую величине 9% от максимального сигнала положительного образца. Кривая флуоресценции не пересекла пороговую линию, что свидетельствовало о том, что в выборке отсутствуют образцы, содержащие серотип-специфическую мишень для 15 ВС серотипа S. pneumoniae. Принадлежность образцов к другим серотипам S. pneumoniae подтверждена анализом данных полногеномного секвенирования с использованием программ «SeroBA» и «PneumoCaT».
Таким образом, заявляемое изобретение позволяет выявлять в биологических образцах ДНК 15 ВС серотипа S. pneumoniae. Синтезированные олигонуклеотиды SEQ ID NO: 1-3 не дают перекрестных реакций с другими протестированными серотипами S. pneumoniae, амлифицируют консервативный для 15 ВС серотипа участок со 100% специфичностью и позволяют определять наличие или отсутствие в образцах биологического материала серотип-специфической мишени для 15 ВС серотипа S. pneumoniae.
| название | год | авторы | номер документа |
|---|---|---|---|
| Мультиплексная ПЦР-смесь для определения серотипов 12FAB, 15BC, 22FA, 8 Streptococcus pneumoniae и способ ее применения | 2021 |
|
RU2787181C1 |
| Олигонуклеотиды для определения 12FAB серотипа Streptococcus pneumoniae | 2021 |
|
RU2795021C1 |
| Олигонуклеотиды для определения 22FA серотипа Streptococcus pneumoniae | 2021 |
|
RU2802203C2 |
| Олигонуклеотиды для определения 8 серотипа Streptococcus pneumoniae | 2021 |
|
RU2802081C2 |
| Набор олигонуклеотидных праймеров и зондов для определения аллей полиморфизма rs55986091 и способ его применения | 2022 |
|
RU2804110C1 |
| Набор олигонуклеотидных праймеров и зондов для видовой дифференциации вируса герпеса человека 6А и вируса герпеса человека 6В и способ его применения | 2022 |
|
RU2806427C1 |
| Способ генотипирования гена TLR8 по полиморфизму rs3764880 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805864C1 |
| Способ генотипирования гена TLR2 по полиморфизму rs5743708 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805861C1 |
| Набор олигонуклеотидных праймеров и зонда для выявления вируса герпеса человека 6В | 2022 |
|
RU2802953C1 |
| Способ генотипирования гена TLR2 по полиморфизму rs3804100 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805860C1 |


Изобретение относится к биотехнологии, в частности к определению 15 ВС серотипа Streptococcus pneumoniae с использованием полимеразной цепной реакции в режиме реального времени (ПЦР-РРВ) и может использоваться для идентификации возбудителей пневмококковых инфекций (ПИ) и оценки эффективности проводимых иммунопрофилактических мероприятий. Олигонуклеотиды для определения 15 ВС серотипа S. pneumoniae - прямой праймер 15BCF, обратный праймер 15BCR, флоуресцентный зонд 15BCZ, представлены уникальными последовательностями SEQ ID NO: 1-3 соответственно. При этом прямой 15BCF и обратный 15BCR праймеры комплементарны участку гена S. pneumoniae 15 ВС серотипа, не имеющим гомологии с фрагментами cps-локуса представителей других серотипов S. pneumoniae. 2 з.п. ф-лы, 3 ил., 1 табл., 4 пр.
1. Набор праймеров и зонда для определения 15BC серотипа Streptococcus pneumoniae методом полимеразной цепной реакции, проводимой в режиме реального времени, и имеющие следующий нуклеотидный состав:
прямой праймер (15BCF) – SEQ ID NO: 1;
обратный праймер (15BCR) – SEQ ID NO: 2;
флоуресцентный зонд (15BCZ) – SEQ ID NO: 3.
2. Набор праймеров и зонда для определения 15BC серотипа Streptococcus pneumoniae по п. 1, где прямой и обратный праймеры комплементарны участку гена S. pneumoniae 15BC серотипа, не имеющим гомологии с фрагментами cps-локуса представителей других серотипов S. pneumoniae.
3. Набор праймеров и зонда для определения 15BC серотипа Streptococcus pneumoniae по п. 1, где флоуресцентный зонд является олигонуклеотидом, содержащим флуорофор R6G и гаситель флуоресценции BHQ1, позволяющим детектировать амплифицированный фрагмент.
| Shakrin N | |||
| et al | |||
| Research Note Evaluation of PCR-based approach for serotype determination of Streptococcus pneumoniae //Tropical biomedicine | |||
| Многоступенчатая активно-реактивная турбина | 1924 |
|
SU2013A1 |
| - Т | |||
| Способ обработки медных солей нафтеновых кислот | 1923 |
|
SU30A1 |
| - No | |||
| Аппарат для очищения воды при помощи химических реактивов | 1917 |
|
SU2A1 |
| - С | |||
| Чемодан с сигнальным замком | 1922 |
|
SU338A1 |
| Миронов К.О | |||
| и др | |||
| Методика ПЦР в режиме реального времени для определения серотипов Streptococcus pneumoniae //Журнал микробиологии, эпидемиологии и | |||
Авторы
Даты
2023-05-02—Публикация
2021-12-24—Подача