Перекрестная ссылка на родственные заявки
Данная заявка испрашивает приоритет по предварительной патентной заявке США №62/275,600, поданной 6 января 2016 года, раскрытие которой включено в данную заявку путем ссылки.
Область техники, к которой относится изобретение Настоящее изобретение в целом касается иммунотерапии и более конкретно рекомбинантных Т-клеточных рецепторов, которые могут наделять Т-клетки способностью к направленному распознаванию опухоли.
Уровень техники
Опухолевые антигенспецифические Т-клетки распознают и уничтожают раковые клетки за счет использования уникального комплекса альфа- и бета-цепи Т-клеточного рецептора (TCR), который является специфичным к комплексу пептид опухолевого антигена/HLA. Чрезвычайно разнообразная альфа/бета последовательность TCR самостоятельно определяет пептидную специфичность, рестрикцию HLA и силу распознавания. Генная инженерия поликлонально расширенных периферических Т-клеток с опухолево-антигенспецифическим TCR генерирует большие количества опухолево-антигенспецифических Т-клеток, которые могут быть использованы в адоптивной Т-клеточной терапии пациентов с раком с применением аутологических генно-сконструированных Т-клеток. В настоящее время, только несколько терапевтических генных продуктов TCR было исследовано в клинических испытаниях, что в значительной мере ограничивает применимость данной мощной терапевтической стратегии к ограниченным пациентам из-за их типов HLA, а также экспрессии антигена в раковых клетках. Таким образом, существует постоянная и неудовлетворенная потребность в улучшенных композициях и способах применения в адоптивной иммунной терапии с использованием рекомбинантных TCR. Настоящее изобретение соответствует этой потребности.
Сущность изобретения
Настоящее изобретение предусматривает в одном аспекте модифицированную Т-клетку человека, содержащую рекомбинантный полинуклеотид, кодирующий альфа-цепь и/или бета-цепь TCR, причем TCR представляет собой один из TCR, обозначенных в данной заявке как AL, KQ, РР, 19305CD8, ВВ, KB, ST, JD, 19305DP, РВ-Р, РВ-Т или РВ13.2, как дополнительно описано ниже. В другом аспекте изобретение включает способ профилактики и/или терапии индивидуума, у которого диагностирован, подозревается наличие или существует риск развития или рецидива рака, причем рак включает раковые клетки, которые экспрессируют NY-ESO-1 и/или его высоко гомологичный антиген LAGE-1, при этом в способе вводят индивидууму модифицированные Т-клетки человека, которые экспрессируют рекомбинантный TCR согласно настоящему изобретению.
В другом аспекте изобретение предусматривает вектор экспрессии, кодирующий TCR, причем TCR содержит альфа-цепь и/или бета-цепь, имеющую последовательность 19305DP AL, KQ, РР, 19305CD8, ВВ, KB, ST, JD, РВ-Р, РВ-Т или РВ13.2, как дополнительно описано ниже.
В другом аспекте изобретение предусматривает библиотеку, содержащую множество векторов экспрессии, причем векторы экспрессии кодируют, по меньшей мере, одну альфа-цепь и/или бета-цепь или их комбинацию, выбранную из группы альфа- и бета-цепей TCR, описанных в данной заявке для TCR 19305DP, AL, KQ, РР, 19305CD8, ВВ, KB, ST, JD, РВ-Р, РВ-Т или РВ13.2, как дополнительно описано ниже. В одном примере, библиотека может дополнительно содержать, по меньшей мере, один вектор экспрессии, кодирующий альфа-цепь, бета-цепь или их комбинацию для TCR JM, 5 В8, SB95, которые также дополнительно описаны ниже.
В другом аспекте изобретение предусматривает способ, в котором выбирают вектор экспрессии из библиотеки согласно настоящему изобретению, при этом выбор основывается, по меньшей мере, частично на типе HLA индивидуума, у которого диагностирован или подозревается наличие NY-ESO-1/LAGE-1 положительного рака, и распределяют выбранный вектор экспрессии для участия в применении при введении вектора экспрессии в иммунные клетки диагностированного индивидуума.
В другом аспекте изобретение предусматривает способ, в котором выбирают вектор экспрессии из библиотеки вектора экспрессии согласно настоящему изобретению, и вводят вектор экспрессии в иммунные клетки, полученные от индивидуума, у которого диагностирован NY-ESO-1/LAGE-1 положительный рак, при этом тип HLA TCR, кодированного вектором экспрессии, сопоставляется с типом HLA индивидуума, причем в способе необязательно вводят иммунные клетки, содержащие вектор экспрессии, индивидууму, который в этом нуждается.
В другом аспекте изобретение предусматривает способ, в котором исследуют образцы от индивидуума для того, чтобы определить имеет ли индивидуум или нет NY-ESO-1/LAGE-1 положительный рак, и после определения того, что индивидуум страдает от NY-ESO-1/LAGE-1 положительного рака, выбирают вектор экспрессии из библиотеки согласно настоящему изобретению, основанный, по меньшей мере, частично на типе HLA индивидуума, и вводят вектор экспрессии в иммунные клетки индивидуума, при этом в способе необязательно дополнительно вводят индивидууму иммунные клетки, содержащие вектор экспрессии.
В другом аспекте изобретение предусматривает компьютеризованные способы выбора и/или извлечения TCR из библиотеки согласно настоящему изобретению для применения в иммунотерапии. В вариантах осуществления, изобретение включает базу данных, содержащую нуклеотидные и/или аминокислотные последовательности TCR, или другие признаки TCR. В некоторых вариантах осуществления, изобретение включает систему, которая содержит процессор, запрограммированный на подбор соответствия TCR из библиотеки согласно настоящему изобретению типу HLA образца.
Краткое описание чертежей
На Фигурах 1A-1D представлены графические изображения векторов согласно настоящему изобретению. Фигура 1А. Для трансдукции Т-клеток, векторы, полученные из вирусов мышиных стволовых клеток (MSCV), широко использовались из-за сильной промоторной активности путем длинных концевых повторов (LTR) MSCV и стабильности in vivo трансгенной экспрессии в гемопоэтических клетках. Поскольку 3'-LTR копируется в 5'-LTR во время интеграции в клетки-хозяева (Т-клетки) и является ответственным за транскрипцию трансгенов, 3'-LTR в плазмидах является важным в экспрессии. С другой стороны, 5'-LTR является ответственным за транскрипцию продуцирования вируса. Схематическое представление для классических векторов, полученных из MSCV (pMIG-II и pMIG-w), показано на Фигуре 1А. Оба вектора имеют LTR, полученный из MSCV, в 5' и 3' сайтах, сигнал упаковки (ψ), множественные сайты клонирования (MCS). Трансген клонируют в множественном сайте клонирования (MCS), за которым следует внутренний сайт связывания рибосомы (IRES) и ген зеленого флуоресцентного белка (GFP) для того, чтобы эффективно обнаруживать трансдуцированные клетки. Вектор pMIG-w имеет дополнительный постатранскрипционный регуляторный элемент вируса гепатита сурков (WRE), который усиливает экспрессию трансгена. Могут быть введены дополнительные модификации, такие как те, которые обнаружены в коммерческом ретровирусном векторе, pDON-5 (Clontech). pDON-5, который получают из вектора вируса лейкоза мыши (MLV), заменяет 5'-LTR на CMV/MLV гибридный LTR для усиления продуцирования вируса посредством сильной промоторной активности CMV в клеточной линии, упаковывающей вирус. Кроме того, частичный интрон из гена фактора элонгации 1α человека может быть введен для того, чтобы обеспечивать акцепторный сайт сплайсинга (SA), который вместе с эндогенным донорным сайтом сплайсинга (SD) индуцирует сплайсинг и усиливает транскрипцию.
Фигура 1В. Для того, чтобы создать ретровирусный вектор, который может продуцировать ретровирус с высоким титром, который индуцирует высокоуровневую трансгенную (TCR) экспрессию в Т-клетках, мы амплифицировали фрагмент ДНК из 5'-LTR в интрон, содержащий акцепторный сайт сплайсинга в pDON-5 плазмиде. Прямой праймер был сконструирован для добавления сайта распознавания рестрикционного фермента SgrAI до 5'-LTR, и обратный праймер был сконструирован для добавления NotI и SalI сайтов после интрона. ПЦР-амплифицированный фрагмент обрабатывали SgrAI и SalI и вставляли в плазмиды pMIG-II и pMIG-w таким образом, чтобы заменить 5'-LTR на GFP.
Фигура 1С. Изображенные плазмиды имеют только сайты распознавания NotI и SalI для клонирования. Использование SalI, известной 6-мерной последовательности, не является предпочтительным, поскольку данная короткая распознаваемая последовательность может появляться в трансгене. Для обеспечения дополнительного сайта распознавания рестрикционного фермента, который, как полагают, отсутствует у большинства трансгенов, которые имеют отношение к настоящему изобретению, мы амплифицировали фрагмент ДНК размером 1,8 тыс. осн. (лишний) с прямым праймером с сайтом рестрикции NotI, и обратным праймером с сайтами PacI-SalI. Амплифицированный фрагмент обрабатывали рестрикционными ферментами NotI и SalI и вставляли в новые плазмиды.
Фигура 1D. Кассета экспрессии TCR была амплифицирована прямым праймером с сайтом рестрикции NotI и обратным праймером с сайтом рестрикционного фермента PacI. Амплифицированную кассету экспрессии обрабатывали рестрикционными ферментами NotI и PacI и вставляли в новые плазмиды.
Фигура 2. Экспрессия 19305DP-TCR на поликлонально активированных Т-клетках. Поликлонально активированные Т-клетки из мононуклеарных клеток периферической крови дважды трансдуцировали ретровирусным вектором, кодирующим 19305DP-TCR, чья вариабельная область бета-цепи представляла собой подтип Vb8. Экспрессию измеряли, используя окрашивание анти-TCR Vb8 антителом вместе с анти-CD4 и анти-CD8 антителами.
Фигура 3. Распознавание раковых клеток 19305DP-TCR-трансдуцированными Т-клетками. 19305DP-TCR трансдуцированные Т-клетки совместно культивировали с NY-ESO-1+SK-MEL-37 и NY-ESO1-SK-MEL-29 меланомными клеточными линиями в течение 6 часов в присутствии Golgi Stop, и продуцирование IFN-g измеряли, используя внутриклеточное IFN-g окрашивание в сочетании с окрашиванием CD8 на поверхности клеток.
Фигура 4, панель А. Ретровирусный вектор, использованный для экспрессии TCR. LTR: длинный концевой повтор; ψ: сигнал упаковки; MCS: множественный сайт клонирования; IRES: внутренний сайт посадки рибосомы; eGFP: усиленный зеленый флуоресцентный белок. Фигура 4, панель В. TCR экспрессирующая кассета. (I) последовательности кДНК, кодирующие β- и α-цепь TCR, соединены с помощью линкера GSG (Gly-Ser-Gly) и последовательности Р2А рибосомального пропуска. (II) последовательности кДНК, кодирующие β- и α-цепь TCR, соединены с помощью сайта распознавания протеазы фурина (RAKR (Arg-Ala-Lys-Arg) ((SEQ ID NO: 59)), линкера SGSG (Ser-Gly-Ser-Gly) (SEQ ID NO: 60), эпитопа V5 и последовательности Р2А рибосомального пропуска.
Фигура 5. Экспрессия на клеточной поверхности молекул CD4 и CD8 на рестрицируемых по HLA-A*02 Т-клеточных клонах. 19305DP-TCR CD4/CD8 двойной положительный Т-клеточный клон и AL CD8 одинарный положительный Т-клеточный клон окрашивали анти-CD4 и анти-CD8 моноклональными антителами и анализировали с использованием проточной цитометрии.
Фигура 6. Влияние блокирующих антител на распознавание мишеней рака. 19305DP-TCR CD4/CD8 двойной положительный Т-клеточный клон и JD CD8 одинарный положительный Т-клеточный клон совместно культивировали с HLA-A*02+NY-ESO-1+ меланомной клеточной линией MZ-MEL-9 в присутствии или в отсутствие указанных антител. Анти-HLA антитела класса I (аАВС) ингибировали распознавание как 19305DP, так и JD. Хотя два анти-CD8 антитела в значительной мере ингибировали распознавание JD, распознавание 19305DP не ингибировалось, показывая CD8-независимое распознавание раковых мишеней. * указывает на статистически значимое (р<0,05) ингибирование.
Фигура 7. Влияние замещения аланином на распознавание пептида NY-ESO-1. Каждый аминокислотный остаток в пептиде NY-ESO-1 (157-170) (DP4 пептид SLLMWITQCFLPVF (SEQ ID NO: 61) был замещен на аланиновый остаток. HLA-A*02-положительную и NY-ESO-1-негативную раковую клетку импульсно приводили или не приводили (-) в контакт с аланин-замещенным (1-14) или природным DP4 пептидом (DP4). Распознавание двойным 19305DP CD4/CD8 положительным Т-клеточным клоном, AL CD8 одинарным положительным Т-клеточным клоном и JD CD8 одинарным положительным Т-клеточным клоном исследовали за счет измерения IFN-g в супернатанте. Аланиновое замещение аминокислот в положениях 2, 3, 4, 5, 6, и 7 (LLMWIT-SEQ ID NO: 62) в значительной мере уменьшало распознавание 19305DP, указывая на то, что данные остатки являются важными для Т-клеточного распознавания. Анализ in silico показал, что не существует никаких известных белков с последовательностью LLMWIT, за исключением NY-ESO-1 и LAGE-1.
Фигура 8. Противоопухолевая активность in vivo 19305DP-TCR-трансдуцированных Т-клеток. NOD/Scid/IL-2Rg-цепь-дефицитным (NSG) мышам подкожно вводили 1×106 клеточной линии HLA-A2+NY-ESO-1+ А375. Мононуклеарные клетки периферической крови (РВМС) были поликлонально активированы и трансдуцированы 19305DP-TCR, AL-TCR или нерелевантным TCR с помощью ретровирусных векторов. На 11-й день, 2,5×105 TCR трансдуцированных Т-клеток вводили внутривенно. Объем опухоли измеряли каждые несколько дней после инъекции Т-клеток цифровым штангенциркулем. 19305-TCR-трансдуцированные Т-клетки в значительной мере ингибировали рост опухоли (Панель А) и продлевали выживание (Панель В). Пять из 6 мышей, получавших 19305DP-TCR-трансдуцированные Т-клетки, не имели опухоли на 40-й день, тогда как 4 из 6 мышей, которым инъекционно вводили AL-TCR-трансдуцированные Т-клетки, имели опухоль в этот момент времени.
Фигура 9. Активность CTL in vitro CD8+ и CD4+ Т-клеток, трансдуцированных 19305DP-TCR. РВМС были истощены по CD4+ или CD8+ Т-клеткам по, используя конъюгированные с биотином анти-CD4 или анти-CD8 антитела с последующим использованием антибиотиновых парамагнитных микрочастиц Dynabeads. Истощенные по CD4 и истощенные по CD8 РВМС были поликлонально активированы и трансдуцированы TCR 19305DP-, AL-TCR или нерелевантным-TCR с помощью ретровирусных векторов. (Панель А) Чистоту CD8+ (истощенных по CD4 клеток) и CD4+ (истощенных по CD8 клеток) Т-клеток, трансдуцированных TCR, определяли с использованием проточной цитометрии перед экспериментом in vivo. (Панель В) Цитотоксическую активность CD8+ или CD4+ Т-клеток, трансдуцированных указанным TCR, против клеточной линии HLA-A2+NY-ESO-1+ Mel624.38, исследовали с использованием кальцеин-АМ анализа после 4 часового инкубирования. (Панель С) Апоптическая клеточная смерть раковых клеток наступала после их совместного культивирования с CD4+ или CD8+ Т-клетками, трансдуцированными указанным TCR. Клеточную линию HLA-A2+NY-ESO-1+А375 (2,5×105) совместно культивировали с 5×105 Т-клетками в течении 20 часов. Апоптическую клеточную смерть определяли путем окрашивания аннексином-V и -PI. В то время, как 19305DP-TCR-трансдуцированные CD4+ Т-клетки показывали недельную цитотоксическую активность по сравнению с CD8+ Т-клетками, используя кратковременный цитотоксический анализ (Панель В), CD4+ Т-клетки индуцировали апоптическую клеточную смерть (Панель С). Смерть раковых клеток не наблюдалась при совместном культивировании с AL-TCR-трансдуцированными CD4+ Т-клетками.
Фигура 10. Противоопухолевая активность CD8+ и CD4+ Т-клеток, трансдуцированных 19305DP-TCR. РВМС были истощены по CD4+ или CD8+ Т-клеткам с использованием конъюгированных с биотином анти-CD4 или анти-CD8 антител, с последующим использованием антибиотиновых парамагнитных микрочастиц Dynabeads. Истощенные по CD4 и истощенные по CD8 РВМС были поликлонально активированы и трансдуцированы 19305DP-TCR, AL-TCR или нерелевантным-TCR с помощью ретровирусных векторов. NOD/Scid/IL-2Rg-цепь-дефицитным (NSG) мышам подкожно вводили 1×106 клеточной линии HLA-A2+NY-ESO-1+ А375. На 11-й день внутривенно инъекционно вводили 0,6×105 TCR трансдуцированных CD8+ Т-клеток (25% от всех РВМС) и 1,9×105 TCR трансдуцированных CD4+ Т-клеток (75% от всех РВМС). Объем опухоли измеряли каждые несколько дней после инъекции Т-клеток, используя цифровой штангенциркуль. Как 19305DP-TCR, так и AL-TCR-трансдуцированные CD8+ Т-клетки контролировали рост опухоли (слева), в то время как только 19305DP-TCR трансдуцированные CD4+ Т-клетки показали регрессию опухоли (справа).
Подробное описание изобретения
Если не указано иное в данной заявке, все технические и научные термины, используемые в настоящем изобретении, имеют такое же значение, которое обычно понимается специалистом в данной области техники, к которой относится настоящее изобретение.
Каждый численный диапазон, указанный в данном описании, включает его верхние и нижние значения, а также каждый более узкий числовой диапазон, попадающий в него, как если бы такие более узкие числовые диапазоны были явно прописаны в данной заявке.
Каждый полинуклеотид, описанный в данной заявке, включает его комплементарную последовательность и его обратную комплементарную последовательность, а также эквиваленты РНК последовательностей ДНК, где каждый Т в последовательности ДНК заменен на U. Все полинуклеотидные последовательности, кодирующие аминокислотные последовательности, описанные в данной заявке, включены в объем настоящего изобретения.
Настоящее изобретение предусматривает композиции и способы профилактики и/или терапии различных видов рака. В целом, раковые заболевания представляют собой заболевания, которые экспрессируют хорошо известный антиген NY-ESO-1/LAGE-1.
Изобретение включает каждый из рекомбинантных TCR, описанных в данной заявке, полинуклеотиды, кодирующие их, векторы экспрессии, содержащие полинуклеотиды, клетки, в которые полинуклеотиды были введены, включая, но необязательно ограничиваясь приведенным, CD4+ T-клетки, CD8+ Т-клетки, природные киллерные Т-клетки, γδ Т-клетки и клетки-предшественники, такие как гемопоэтические стволовые клетки. Как используется в данной заявке, "рекомбинантный TCR" означает TCR, который экспрессируется из полинуклеотида, который был введен в клетку, что означает, что до первоначального введения полинуклеотида TCR не кодировался хромосомной последовательностью или другим полинуклеотидом в клетке.
В вариантах осуществления, клетки, в которые вводятся полинуклеотиды, представляют собой лимфоидные клетки-предшественники, незрелые тимоциты (двойные негативные CD4-CD8-) или двойные положительные тимоциты (CD4+CD8+). В вариантах осуществления, клетки-предшественники содержат маркеры, такие как CD34, CD117 (c-kit) и CD90 (Thy-1). Изобретение включает способы получения TCR, способы модифицирования клеток таким образом, что они экспрессируют TCR, модифицированные клетки и способы применения модифицированных клеток для противораковых подходов. Кроме того, включены библиотеки различных TCR.
В конкретных вариантах осуществления изобретение включает способ профилактики и/или терапии индивидуума, у которого диагностирован, подозревается наличие или существует риск развития или рецидива рака, причем рак включает раковые клетки, которые экспрессируют антиген NY-ESO-1/LAGE-1. Данный подход включает, в одном аспекте, введение индивидууму модифицированных клеток человека, содержащих рекомбинантный полинуклеотид, кодирующий TCR согласно настоящему изобретению. Модифицированные клетки человека, такие как модифицированные Т-клетки, являются улучшенными по сравнению с их немодифицированными Т-клеточными аналогами касательно их способности бороться с раком. Таким образом, в вариантах осуществления, реализуемый на практике способ согласно настоящему изобретению в результате приводит к терапевтическому ответу, который может включать, но не обязательно ограничивается приведенным, замедление скорости роста раковых клеток и/или опухолей, уменьшение объема опухоли и/или уменьшение увеличения скорости увеличения объема опухоли, уничтожение раковых клеток, продление срока жизни индивидуума, у которого диагностирован рак, как более полно описано в данной заявке, и другие параметры, которые будут очевидны для специалистов в области лечения рака.
Эффект практических вариантов осуществления настоящего изобретения может быть сравним с любым подходящим эталоном, таким как положительный или отрицательный контроль. В вариантах осуществления эффект может сравниваться с эталонным значением, полученным, например, от контрольных клеток экспрессирующих TCR с известными эффектами, или TCR, который не является специфичным для конкретного рассматриваемого антигена, или TCR, который несогласован по отношению к типу HLA и/или типу Т лимфоцитов, или любое другое приемлемое эталонное значение, которое будет очевидным для специалиста в данной области техники, когда предусматривается преимущество настоящего изобретения. В вариантах осуществления приемлемое эталонное значение включает известное значение или диапазон значений. В вариантах осуществления, эталонное значение включает статистическое значение, такое как площадь под кривой, или другая площадь, или наносится на график, и/или получается из повторяемых измерений.
Различные и неограничивающие варианты осуществления изобретения демонстрируются с использованием рестрицируемых по HLA-I и HLA-II TCR.
TCR, рестрицируемые по HLA-I
В одном аспекте изобретение охватывает новые TCR, которые являются специфичными для антигена NY-ESO-1, как представлено в контексте HLA класса I. Конкретные примеры α-цепи и β-цепи рестрицируемых по HLA-I TCR и полинуклеотидных последовательностей, кодирующих их, описаны дополнительно ниже в виде "19305DP", "AL", "KQ", "РР", "19305CD8", "ВВ", "KB", "ST" и "JD". Данные TCR являются специфичными для полученных из NY-ESO-1/LAGE-1 пептидов, представленных различными типами HLA класса I, включая HLA-A*02; В*27; В*35; Cw*03; и Cw*15.
TCR, рестрицируемые по HLA-II
В другом аспекте изобретение охватывает новые TCR, которые являются специфичными для антигена NY-ESO-1, как представлено в контексте HLA класса II. Конкретные примеры α-цепи и β-цепи рестрицируемых по HLA-II TCR и полинуклеотидных последовательностей, кодирующих их, описаны дополнительно ниже в виде "РВ-Р", "РВ-Т" и "РВ13.2". Данные TCR являются специфичными для полученных из NY-ESO-1/LAGE-1 пептидов, представленных различными типами HLA класса II, включая HLA-DRB1*04 и DRB1*07. В некоторых вариантах осуществления данные TCR наделяют Т-клетки, которые их экспрессируют, способностью непосредственного распознавания опухолей и/или раковых клеток, которые экспрессируют антиген NY-ESO-1/LAGE-1.
В некоторых аспектах, клетки, содержащие рекомбинантный TCR согласно настоящему изобретению, которые вводят индивидууму, представляют собой аллогенные, изогенные или аутологические клетки. Таким образом, в одном варианте осуществления, клетки получают от первого индивидуума, модифицируют и вводят второму индивидууму, который в этом нуждается. В другом варианте осуществления клетки удаляют у индивидуума перед модифицированием, модифицируют для того, чтобы они экспрессировали рекомбинантный TCR, и вводят обратно тому же индивидууму. В некоторых вариантах осуществления, клетки, которые модифицируют в соответствии с настоящим изобретением, включают популяцию иммунных клеток, которая обогащена, содержит или состоит из определенного типа иммунных клеток. В некоторых аспектах клетки представляют собой CD4+ Т-клетки или представляют собой CD8+ Т-клетки. В одном аспекте, клетки, в которые вводят один или несколько векторов экспрессии согласно настоящему изобретению, содержат смесь из иммунных клеток, таких как CD4+ и CD8+ Т-клетки, и/или могут содержать мононуклеарные клетки периферической крови (РВМС). В одном из подходов один или несколько векторов экспрессии, которые самостоятельно или вместе кодируют TCR 19305DP, вводят в смесь иммунных клеток. В некоторых вариантах осуществления, Т-клетки способны к непосредственному распознаванию раковых клеток, экспрессирующих антиген NY-ESO-1/LAGE-1. В вариантах осуществления непосредственное распознавание включает рестрицируемое по HLA класса II связывание TCR с антигеном NY-ESO-1/LAGE-1, экспрессируемым раковыми клетками.
Касательно 19305DP TCR, настоящее изобретение демонстрирует определенные характеристики его экспрессии и функции. Например, на Фигурах 5-7 представлены характеристики родительского клона 19305DP. В частности, на Фигуре 5 приведены данные о CD4/CD8 двойных положительных характеристиках. На Фигуре 6 представлена демонстрация CD8-независимого распознавания, которое указывает на высокое распознавание аффинности посредством TCR. На Фигуре 7 представлена демонстрация строгой специфичности последовательности NY-ESO-1/LAGE-1, которая не поддерживает перекрестной реактивности других антигенов человека. На Фигурах 8-10 представлены результаты, демонстрирующие противоопухолевые эффекты Т-клеток человека, трансдуцированных 19305DP-TCR, по сравнению с AL-TCR, который имеет такую же HLA-A*02:01-рестрикцию и NY-ESO-1-специфичность, но получен из CD8 одинарного положительного клона Т-клетки. На Фигуре 8 представлена демонстрация ингибирования роста in vivo NY-ESO-1 и HLA-А*02:01-экспрессирующей меланомы человека за счет мононуклеарных клеток периферической крови человека, содержащих как CD4+, так и CD8+ Т-клетки, которые были трансдуцированы 19305DP или AL TCR-экспрессирующими ретровирусными векторами, что демонстрирует превосходный противоопухолевый эффект 19305DP-TCR-трансдуцированных РВМС.На Фигуре 9 представлена демонстрация способности к уничтожению опухоли in vitro 19305DP и AL TCR-экспрессирующих CD4+ и CD8+ Т-клеток, демонстрируя то, что как CD4+, так и CD8+ Т-клетки эффективно уничтожают NY-ESO-1+HLA-A*02:01+ раковую мишень, когда они были трансдуцированы 19305DP-TCR, в тоже время только CD8+ Т-клетки уничтожали мишень, когда они были трансдуцированы AL-TCR. На Фигуре 10 представлены данные, демонстрирующие ингибирование роста опухоли in vivo 19305DP и AL TCR-экспрессирующими CD4+и CD8+Т-клетками, демонстрирующие, что как CD4+, так и CD8+ Т-клетки атакуют раковые мишени NY-ESO-1+HLA-A*02:01+, когда они были трансдуцированы 19305DP-TCR, в тоже время только CD8+ Т-клетки показали ингибирование роста опухоли, когда они были трансдуцированы AL-TCR. Таким образом, в некоторых вариантах осуществления изобретение касается способности трансдуцировать или CD4+ Т-клетки, или CD8+ Т-клетки, или так называемые двойные положительные ("DP") Т-клетки, используя рекомбинантные TCR и модифицированные клетки согласно настоящему изобретению, такие как клетки, модифицированные для того, чтобы экспрессировать TCR 19305DP.
Касательно DP Т-клеток, то в данной области известно, что они существуют на стадии развития тимуса. CD4+/CD8+ двойные положительные Т-клетки дифференцируются по клеточной линии CD4 или клеточной линии CD8 с помощью способа, известного как тимусный положительный выбор, и пути дифференциации CD4 и CD8, как полагают, являются в целом взаимоисключающими преобразованиями. Тем не менее, зрелые CD4+/CD8+DP Т-клетки были описаны в крови и периферических лимфоидных тканях, и наблюдались при определенных расстройствах, включая рак - хотя и с низкой частотой. Не намереваясь ограничиваться какой-либо конкретной теорией, считают, что настоящее изобретение обеспечивает первое описание TCR, полученного из DP Т-клетки, и продуцирование такого TCR рекомбинантно для того, чтобы продемонстрировать его полезность в адаптивных иммунологических подходах, как продемонстрировано в Примерах и на Фигурах в соответствии с настоящим изобретением. В некоторых вариантах осуществления, изобретение предусматривает смеси клеток, экспрессирующих TCR, или клеток, экспрессирующих больше, чем один TCR, описанный в данной заявке, который является специфичным для разных антигенов рака, таким образом, обеспечивая клеточные популяции, которые могут рассматриваться поливалентными по отношению к TCR.
TCR, предусматриваемый изобретением в некоторых примерах, является способным распознавать NY-ESO-1; 157-170, который представляет собой антиген, состоящий из аминокислотной последовательности SLLMWITQCFLPVF (SEQ ID NO: 63), или является способным распознавать NY-ESO-1;95-106, который представляет собой антиген, состоящий из аминокислотной последовательности PFATPMEAELAR (SEQ ID NO: 64).
В некоторых вариантах осуществления, клетки, предусматриваемые изобретением, представляют собой сконструированные CD8+ Т-клетки, экспрессирующие TCR согласно настоящему изобретению, которые могут непосредственно распознавать NY-ESO-1/LAGE-1+ раковые клетки, и CD4+ Т-клетки, которые способны распознавать данные антигены NY-ESO-1/LAGE-1 через TCR, которые взаимодействуют с антигеном в сочетании с молекулами HLA класса II, при этом молекулы HLA класса II воспроизводятся опухолевыми клетками.
Изобретение включает каждую полинуклеотидную последовательность, которая кодирует один или несколько полипептидов TCR согласно изобретению и раскрывается в данной заявке, включая ДНК и РНК последовательности, и включая выделенные и/или рекомбинантные полинуклеотиды, содержащие и/или состоящие из таких последовательностей. Изобретение также включает клетки, которые содержат рекомбинантные полинуклеотиды. Клетки могут представлять собой выделенные клетки, клетки, выращенные и/или распространенные, и/или поддерживаемые в культуре, и могут представлять собой прокариотические или эукариотические клетки. Прокариотические или эукариотические клеточные культуры могут быть использованы, например, для размножения или амплификации векторов экспрессии TCR согласно изобретению. В вариантах осуществления, клетки могут содержать упаковывающие плазмиды, которые, например, обеспечивают некоторые или все белки, используемые для транскрипции и упаковки копии РНК экспрессирующего конструкта в рекомбинантные вирусные частицы, такие как псевдовирусные частицы. В вариантах осуществления, векторы экспрессии временно или стабильно вводятся в клетки. В вариантах осуществления, векторы экспрессии интегрируются в хромосому клеток, используемых для их продуцирования. В вариантах осуществления, полинуклеотиды, кодирующие TCR, которые вводятся в клетки посредством вектора экспрессии, такие как вирусная частица, являются интегрированными в одну или несколько хромосом клеток. Такие клетки могут быть использованы для распространения, или они могут представлять собой клетки, которые используются для терапевтических и/или профилактических подходов. Эукариотические клетки включают CD4+ Т-клетки, CD8+ Т-клетки, природные киллерные Т-клетки, γδ Т-клетки, и их клетки-предшественники, в которые был введен конструкт экспрессии TCR согласно изобретению. CD4+ Т-клетки могут быть из любого источника, включая, но не ограничиваясь приведенным, субъекта, такого как человек, который может или не может быть конечным получателем CD4+ Т-клеток, CD8+ Т-клеток или их комбинаций, как только они были сконструированы для экспрессии нового TCR в соответствии с настоящим изобретением.
Векторы экспрессии для применения согласно вариантам осуществления настоящего изобретения могут представлять собой любой приемлемый вектор экспрессии. В вариантах осуществления, вектор экспрессии включает модифицированный вирусный полинуклеотид, такой как из аденовируса, герпесвируса или ретровируса, такой как лентивирусный вектор. Вектор экспрессии не ограничивается рекомбинантными вирусами и включает невирусные векторы, такие как ДНК-плазмиды и in vitro транскрибируемая мРНК.
Касательно полипептидов, которые кодируются полинуклеотидами/векторами экспрессии, описанными выше, в некоторых аспектах изобретение предусматривает функциональные TCR и векторы экспрессии, кодирующие их, при этом функциональный TCR, который включает α- и β-цепь TCR, причем две цепи присутствуют в физической ассоциации друг с другом (например, в комплексе) и нековалентно соединены друг с другом, или две цепи представляют собой различные полипептиды, но при этом ковалентно соединенные друг с другом, например, посредством дисульфидной или другой ковалентной связи, которая не является пептидной связью. Другие приемлемые связи могут включать, например, замещенный или незамещенный полиалкиленгликоль, и комбинации этиленгликоля и пропиленгликоля в форме, например, сополимеров. В других вариантах осуществления, два полипептида, которые составляют α- и β-цепь TCR, оба могут быть включены в один полипептид, такой как слитый белок. В некоторых вариантах осуществления, слитый белок содержит аминокислотную последовательность α-цепи TCR и аминокислотную последовательность β-цепи TCR, которые были транслированы из одной и той же открытой рамки считывания (ORF), или отдельных ORF, или ORF, которая содержит сигнал, который в результате приводит к прерывистой трансляции. В одном варианте осуществления, ORF содержит Р2А-опосредованный сайт пропуска трансляции, расположенный между α- и β-цепью TCR. Конструкты для получения белков, содержащих Р2А (также называемые как пептид 2А-связанные мультицистронные векторы), известны в данной области техники. (См., например, Gene Transfer: Delivery and Expression of DNA and RNA, A Laboratory Manual, (2007), Friedman et al., International Standard Book Number (ISBN) 978-087969765-5. Кратко, последовательности пептида 2A, когда включены между кодирующими областями, позволяют стехиометрическое продуцирование дискретных белковых продуктов в пределах одного вектора через новое расщепление, которое происходит в последовательности пептида 2А. Последовательности пептида 2А, как правило, представляют собой короткую последовательность, содержащую 18-22 аминокислоты, и могут включать различные аминоконцевые последовательности. Таким образом, в одном варианте осуществления, слитый белок согласно изобретению включает аминокислотную последовательность Р2А. В вариантах осуществления, слитый белок согласно изобретению может включать линкерную последовательность между α- и β-цепями TCR. В некоторых вариантах осуществления, линкерная последовательность может содержать GSG (Gly-Ser-Gly) линкер или SGSG (Ser-Gly-Ser-Gly) (SEQ ID NO: 59) линкер. В некоторых вариантах осуществления, α- и β-цепи TCR связаны друг с другом аминокислотной последовательностью, которая включает сайт распознавания протеазы фурина, такой как сайт RAKR (Arg-Ala-Lys-Arg) (SEQ ID NO: 60).
В одном варианте осуществления, конструкт экспрессии, который кодирует TCR, также может кодировать дополнительные полинуклеотиды. Дополнительный полинуклеотид может быть таким, что он позволяет идентифицировать экспрессирующие TCR клетки, например, путем кодирования детектируемого маркера, такого как флуоресцентный или люминесцентный белок. Дополнительный полинуклеотид может быть таким, что он кодирует элемент, который позволяет селективно удалять экспрессирующие TCR клетки, например, ген тимидинкиназы. В вариантах осуществления дополнительные полинуклеотиды могут быть такими, что они способствуют ингибированию экспрессии эндогенно кодированных TCR. В варианте осуществления конструкт экспрессии, который кодирует TCR, также кодирует полинуклеотид, который может облегчить опосредованную РНК-интерференцией понижающую регуляцию одного или нескольких эндогенных TCR. Например, см. Okamoto S, et al. (2009) Cancer Research, 69:9003-9011, и Okamoto S, et al. (2012). Molecular Therapy-Nucleic Acids, 1, ебЗ. В варианте осуществления, конструкт экспрессии, который кодирует TCR, может кодировать кшРНК или миРНК, нацеленные на эндогенно кодированный TCR. В альтернативном варианте осуществления, может быть использован второй, отличающийся конструкт экспрессии, который кодирует полинуклеотид для применения в понижающе регулирующем эндогенном продуцировании TCR.
В некоторых подходах различные цепи TCR могут быть экспрессированы из конструкта экспрессии таким образом, что β-цепь ориентирована N-терминально по отношению к α-цепи, и таким образом TCR согласно изобретению также могут содержать данную ориентацию цепи, или другие ориентации. В альтернативных вариантах осуществления белки α- и β-цепей TCR могут быть экспрессированы из различных векторов экспрессии, введенных в одну и ту же клетку. В некоторых вариантах осуществления, мРНК, кодирующая TCR, может быть использована в качестве альтернативы векторам экспрессии.
Касательно применения сконструированных CD4+ Т-клеток, CD8+ Т-клеток и их комбинаций, способ, как правило, включает введение эффективного количества (как правило, 1010 клеток путем внутривенных или внутрибрюшинных инъекций) композиции, содержащей CD4+ Т-клетки, индивидууму, который нуждается в этом. Индивидуум, который нуждается в этом, в различных вариантах осуществления, представляет собой индивидуум, который имеет или у которого подозревается наличие, или существует риск развития рака, который характеризуется злокачественными клетками, которые экспрессируют NY-ESO-1/LAGE-1. Как хорошо известно в данной области, NY-ESO-1/LAGE-1 экспрессируется различными раковыми клетками и типами опухолей. В частности и в качестве неограничивающих примеров, такие типы рака включают рак мочевого пузыря, головного мозга, молочной железы, яичника, немелкоклеточный рак легкого, миелому, рак предстательной железы, саркому и меланому. Конкретные варианты осуществления включают, но не ограничиваются приведенным, липосаркомы и внутрипеченочную холягиокарциному. У индивидуума могут быть ранние стадии или прогрессирующие формы любого из данных видов рака, или может быть в стадии ремиссии любой из данных видов рака. В одном варианте осуществления, индивидуум, которому вводится композиция согласно изобретению, подвергается риску рецидива для любого вида рака, который экспрессирует NY-ESO-1. В некоторых вариантах осуществления, индивидуум имеет или у него подозревается наличие, или существует риск развития или рецидива опухоли, содержащей клетки, которые экспрессируют белок, содержащий аминокислотные последовательности, определенные NY-ESO-1:157-170 и/или NY-ESO-1:95-106. В вариантах осуществления, изобретение включает рекомбинантные TCR, которые являются специфичными для пептидных фрагментов NY-ESO-1, которые имеют в длину от 15 до 24 аминокислотных остатков, причем такие пептиды представлены в комплексе с HLA-II. В вариантах осуществления, изобретение включает рекомбинантные TCR, которые являются специфичными для пептидов, которые находятся в комплексе с HLA-I или HLA-II, причем пептиды содержат или состоят из аминокислотных последовательностей NY-ESO-1:157-170 и/или NY-ESO-1:95-106.
Нуклеотидные и аминокислотные последовательности, представленные ниже, представляют собой те, которые используются для демонстрации изобретения. Как описано выше, изобретение включает любую и все полинуклеотидные последовательности, кодирующие аминокислотные последовательности конструктов TCR, описанных в данной заявке. Кроме того, рассматриваются вариации аминокислотных последовательностей в TCR, при условии, что они не оказывают отрицательного влияния на функцию TCR. В различных вариантах осуществления, TCR, содержащий одно или несколько аминокислотных изменений по сравнению с последовательностями, представленными в данной заявке, будет содержать консервативные аминокислотные замещения или другие замещения, инсерции или делеции, при условии, что клетки, экспрессирующие рекомбинантные TCR согласно изобретению, могут непосредственно и специфически распознавать опухолевые клетки, которые экспрессируют NY-ESO-1/LAGE-1, при этом это распознавание зависит от экспрессии NY-ESO-1/LAGE-1 и презентации пептидов, процессированных из него при рестрикции по HLA класса II опухолевыми клетками. В вариантах осуществления, TCR согласно настоящему изобретению содержит любую аминокислотную последовательность, которая облегчает непосредственное распознавание опухолевого антигена на опухолевых клетках, без участия клеток, представляющих антиген. В вариантах осуществления, аминокислотная последовательность TCR, предусматриваемая настоящим изобретением, является, по меньшей мере, на 95%, 96%, 97%, 98% или 99% подобной аминокислотным последовательностям, представленным в перечне последовательностей, который является частью настоящего изобретения. В различных вариантах осуществления, любой TCR согласно изобретению может иметь значение Koff для его родственного эпитопа, как определено в данной заявке, которое по существу является таким же, как Koff для родственного эпитопа, продемонстрированного TCR, встречающегося в природе TCR для такого же эпитопа. В вариантах осуществления, аминокислотные последовательности TCR могут включать изменения в их константной области. В этой связи в данной области техники известно, что, в основном, константная область TCR по существу не способствует распознаванию антигена. Например, возможным является заместить часть константной области TCR человека на мышиную последовательность и сохранить функцию TCR. (См., например, Goff SL et al. (2010) Cancer Immunology, Immunotherapy, 59: 1551-1560). Таким образом, предполагаются различные модификации последовательностей TCR, раскрытых в данной заявке, и они могут включать, но не ограничиваются приведенным, изменения, которые улучшают специфическое спаривание цепей или улучшают более сильную ассоциацию с сигнальными белками Т-клеток комплекса CD3, или ингибируют образование димеров между эндогенными и введенными TCR. В вариантах осуществления, аминокислотные изменения могут присутствовать в области CDR, такой как область CDR3, включая, но необязательно ограничиваясь приведенным, замещения одной, двух, трех или более аминокислот в последовательности CDR3. В вариантах осуществления, аминокислотные изменения не оказывают влияния на функцию TCR. Зрелым белкам TCR предшествуют аминокислотные последовательности, называемые сигнальным пептидом или лидерным пептидом, которые направляют вновь синтезированные белки TCR на секреторный путь. Сигнальный пептид удаляют из зрелого белка TCR перед экспрессией на клеточной поверхности. Таким образом, замещение сигнального пептида на другую природную или искусственную последовательность не изменяет функцию зрелого TCR. Таким образом, предполагаются различные модификации сигнальных пептидных последовательностей, раскрытых в данной заявке, включая, но не ограничиваясь приведенным, делецию или изменение некоторых или всех из аминокислот, находящихся в сигнальном пептиде, или замещение всех или некоторых из аминокислот на другие аминокислоты и/или полипептидные последовательности, примеры которых будут очевидны специалистам в данной области техники, учитывая преимущество настоящего изобретения. В некоторых аспектах, изобретение включает векторы экспрессии и другие полинуклеотиды, кодирующие один или несколько гипервариабельных TCR или комплементарных определяющих областей (CDR) из альфа-цепи, бета-цепи TCR или их комбинации. В некоторых вариантах осуществления кодируется только одна CDR или кодируются только две CDR, или кодируются только три CDR, или кодируются комбинации из только определенных CDR из альфа- и бета-цепей TCR, и все такие комбинации сегментов CDR TCR и полинуклеотидов, кодирующих их из альфа- и бета-цепей TCR, описанных в данной заявке, охватываются настоящим изобретением. Специалисты в данной области техники будут способны распознавать сегменты CDR TCR каждой из аминокислотних последовательностей TCR, представленных в данной заявке. Библиотеки
Изобретение включает множество векторов экспрессии, кодирующих TCR, то есть библиотеку содержащую множество различных векторов экспрессии, кодирующих различные TCR, при этом, по меньшей мере, один член библиотеки кодирует новую α-цепь и/или новую β-цепь TCR согласно настоящему изобретению. Таким образом, по меньшей мере, один член библиотеки может быть выбран из векторов экспрессии, которые кодируют α-цепь и/или β-цепь, по меньшей мере, одного из рестрицируемых по HLA-I TCR, упоминаемых в данной заявке как AL, KQ, РР, 19305CD8, ВВ, KB, ST, JD и 19305DP, и, по меньшей мере, член библиотеки может быть выбран из векторов экспрессии, которые кодируют α-цепь и/или β-цепь, по меньшей мере, одного из новых рестрицируемых по HLA-II TCR, описанных в данной заявке РВ-Р, РВ-Т, РВ13.2. Комбинации различных векторов экспрессии, кодирующих данные рестрицируемые по HLA-I и HLA-II TCR, включены в изобретение.
В одном неограничивающем примере библиотека согласно настоящему изобретению содержит вектор экспрессии, кодирующий TCR, описанный в данной заявке как 19305DP, который представляет собой рестрицируемый по HLA-A*02 TCR и является функциональным в как CD4+, так и CD8+ Т-клетках. Данный TCR был первоначально получен из уникальных опухолево-антигенспецифических Т-клеток, которые были CD4+CD8+ двойными-положительными, и это, как считается, является первым описанием такого TCR. Гены TCR AL, KQ, РР, 19305CD8, ВВ, KB, ST и JD были первоначально получены из CD8+ одинарных положительных Т-клеток, а РВ-Р, РВ-Т и РВ13.2 были из CD4+ одинарных положительных Т-клеток.
В некоторых аспектах, кроме того, по меньшей мере, один новый TCR/вектор экспрессии, описанный в данной заявке, библиотека, предусматриваемая настоящим изобретением, может дополнительно содержать векторы экспрессии, кодирующие рестрицируемые по HLA класса II TCR, выбранные из TCR, описанных ниже как "JM", "5 В8", которые представляют собой рестрицируемые по HLA DPB1*04 и рестрицируемые по DRB1*01 "SB95". Данные конструкты описаны в PCT/US14/25673, опубликованной как WO/2014/160030, из которой описание TCR, векторов экспрессии, кодирующих TCR, и способов получения и применения TCR и векторов экспрессии включено в данную заявку путем ссылки. TCR JM, 5 В8 и SB95 были получены из NY-ESO-1 положительных индивидуумов, и данные TCR предоставляют Т-клеткам, включая CD4+ Т-клетки, способность непосредственного распознавания NY-ESO-1+ раковых клеток, как дополнительно описано ниже, включая, но необязательно ограничиваясь приведенным, РВ-Р, РВ-Т и РВ13.2. Таким образом, данные TCR наделяют Т-клетки способностью непосредственно распознавать раковую клетку, экспрессирующую антиген NY-ESO-1/LAGE-1, причем непосредственное распознавание раковой клетки включает рестрицируемое по человеческим лейкоцитарным антигенам HLA класса II связывание TCR с антигеном NY-ESO-1/LAGE-1, экспрессированным раковой клеткой.
В различных вариантах осуществления библиотека векторов экспрессии согласно настоящему изобретению кодирует разнообразные TCR. В некоторых аспектах, векторы экспрессии не содержат какую-либо фаговую или фагемидную ДНК, и/или никакой(ие) полипептид(ы) TCR не содержит(ат) какой-либо фаговый или фагемидный белок. В вариантах осуществления TCR не содержат какой-либо фаговый или фагемидный белок и, таким образом, не являются компонентами, например, библиотеки фагового дисплея TCR.
В некоторых примерах векторы экспрессии в библиотеке векторов экспрессии TCR согласно настоящему изобретению кодируют множество TCR, так что клетки, экспрессирующие TCR, могут функционировать у разных пациентов с различными типами HLA класса I, типами HLA класса II и/или их комбинациями.
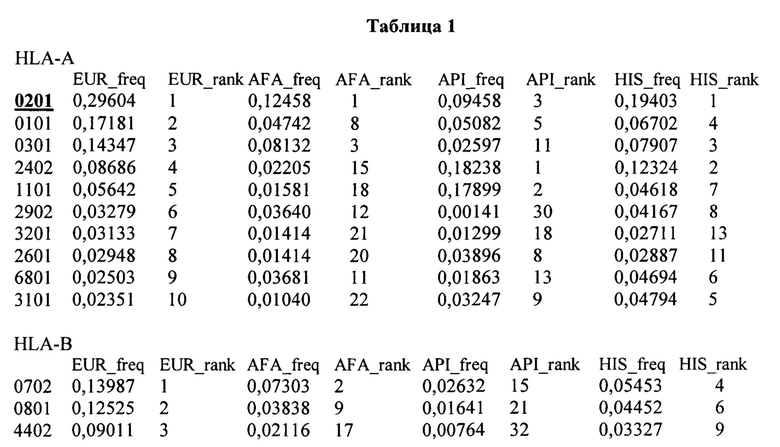
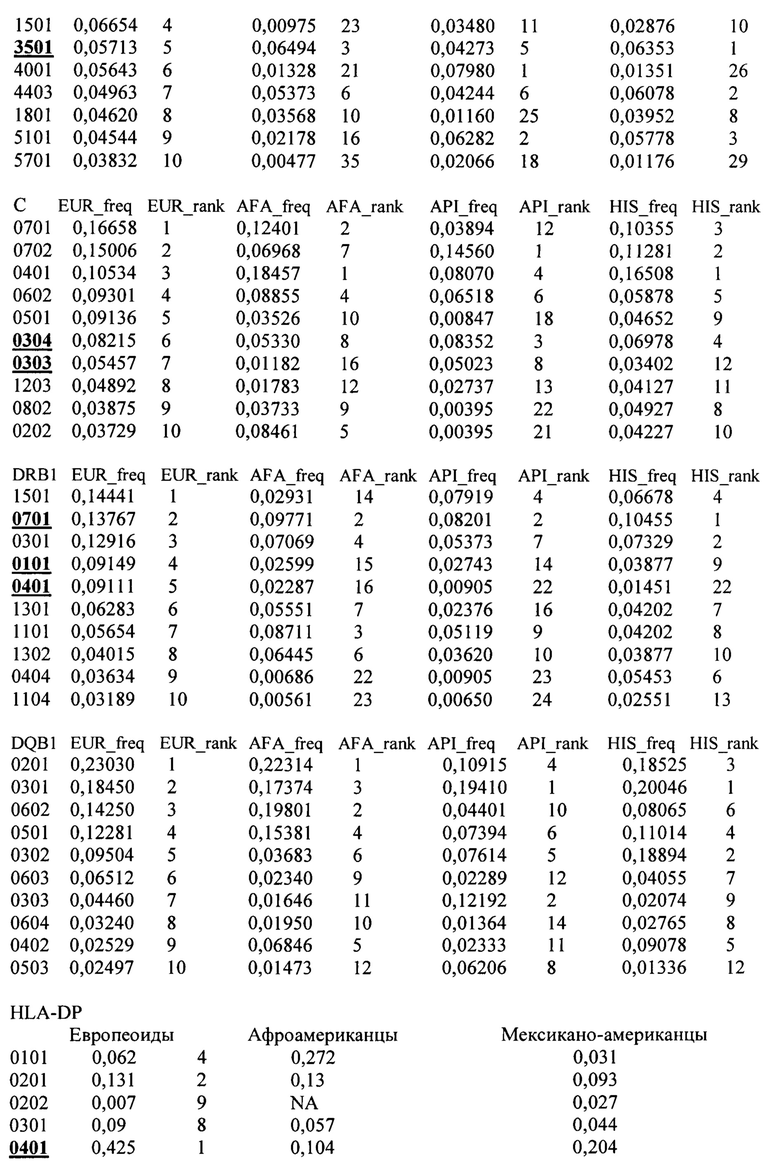
В некоторых вариантах осуществления рестрицируемые по HLA класса I TCR, кодируемыее библиотекой векторов экспрессии согласно настоящему изобретению, способны функционировать у пациентов с типом HLA класса I, выбранным из аллеля, охватываемого HLA-A, -В, -С, и их комбинацией. В некоторых вариантах осуществления, библиотека является достаточно разнообразной для того, чтобы быть приемлемой для применения в терапии рака у, по меньшей мере, 50% европейской расы населения США на момент подачи этой заявки или патента. В некоторых аспектах, библиотека согласно настоящему изобретению является приемлемой для применения в терапии рака на основе рестрицируемых по HLA-класса I (А*02/В*35/С*04) TCR, которая без намерения быть связанной какой-либо конкретной теорией, как полагают, включает 67% типов HLA европейской расы населения США на момент подачи этой заявки или патента, и рестрицируемые по HLA класса II (DR*01/DR*04/DR*07/DP*04) TCR, которые без намерения быть связанными какой-либо конкретной теорией, как полагают, включает 87% типов HLA европейской расы населения США на момент подачи этой заявки или патента. В некоторых вариантах осуществления библиотека TCR согласно настоящему изобретению включает TCR, специфичные для 10 наиболее часто встречающихся типов HLA у европейской расы населения США на момент подачи этой заявки или патента, и, таким образом, может быть приемлемой для применения в терапии рака, по меньшей мере, у 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, или больше населения. В некоторых вариантах осуществления, изобретение предусматривает библиотеку TCR, которая ограничена множеством NY-ESO-1 специфичных TCR, которые подвергаются рестрикции по типам HLA, представленным в Таблице 1. В вариантах осуществления, библиотека содержит TCR, которые подвергаются рестрикции по одному или комбинации подчеркнутых типов HLA в Таблице 1. В некоторых аспектах изобретение включает библиотеку из 2-3 тысяч различных векторов экспрессии, кодирующих различные TCR.
В некоторых вариантах осуществления, векторы экспрессии в библиотеке векторов экспрессии TCR согласно настоящему изобретению кодируют диапазон TCR, таким образом, что клетки, экспрессирующие TCR, могут функционировать у разных пациентов с типом HLA класса И, выбранным из аллеля, охватываемого HLA- DP, -DM, -DOA, -DOB, -DQ, -DR и их комбинации.
В вариантах осуществления, библиотека векторов экспрессии согласно настоящему изобретению содержит, по меньшей мере, два вектора экспрессии, кодирующих, по меньшей мере, два различных TCR, по меньшей мере, один из которых выбирают из 19305DP, AL, KQ, РР, 19305CD8, ВВ, KB, ST, JD, РВ-Р, РВ-Т и РВ13.2. В варианте осуществления библиотека векторов экспрессии согласно настоящему изобретению включает вектор экспрессии, кодирующий альфа-цепь, бета-цепь, или как альфа, так и бета-цепи 19305DP.
В некоторых вариантах осуществления, изобретение включает библиотеку различных векторов экспрессии. В одном примере, каждый вектор экспрессии может содержаться в виде выделенного препарата ДНК, или может поддерживаться, например, в культуре клеток. Такие композиции могут храниться, например, в отдельных герметичных контейнерах, таких как стеклянные или пластиковые флаконы, пробирки Эппендорфа и тому подобное, и могут храниться при пониженной температуре, такой как температура, равная нулю градусам С, или ниже. Каждый отдельный контейнер или место, где хранится контейнер, может включать указание вектора(ов) экспрессии в контейнере. Такие указания могут включать, но не ограничиваются приведенным, материал, воспринимаемый человеком или машиной, как например отпечатанную этикетку, штрих-код, QR код, и тому подобное, или любое другое указание, которое является полезным для идентификации содержимого/местоположения вектора экспрессии, и которое может быть использовано для извлечения TCR для применения в способе согласно настоящему изобретению. В некоторых аспектах, изобретение включает множество отдельных контейнеров, содержащих различные TCR, причем каждый контейнер индексируется, и при этом указания контейнеров хранятся в базе данных. База данных может быть оцифрована и может быть адаптирована таким образом, чтобы она была интегрирована в программное обеспечение. В некоторых аспектах, изобретение предусматривает компьютерный способ для выбора TCR из библиотеки согласно настоящему изобретению, причем способ включает использование процессора для проверки на совпадение вводимого типа HLA индивидуума, или образца, или другой информации HLA с TCR в библиотеке, который совместим с указанным типом HLA. В некоторых вариантах реализациях, изобретение может исключать компьютерные подходы, которые включают сигналы, несущие волны или транзиторные сигналы.
В одном аспекте изобретение включает систему, содержащую библиотеку TCR, и базу данных, содержащую указания нуклеотида и/или последовательностей, и/или типа HLA TCR, базу данных, сообщающуюся с процессором, причем процессор запрограммирован на выбор и/или обозначение приемлемого TCR в библиотеке, который соответствует типу HLA образца. Система может дополнительно содержать устройство для извлечения контейнера, который содержит подобранный TCR, включая, но не обязательно, роботизированное устройство, которое может, например, быть направлено на указание приемлемого TCR, и может выбирать и/или извлекать указанный TCR из библиотеки. В другом аспекте изобретение предусматривает машиночитаемый носитель, содержащий базу данных, заполненную информацией о библиотеке TCR согласно настоящему изобретению. В варианте осуществления, изобретение включает машиночитаемый носитель, содержащий набор команд для компьютера для выбора TCR из библиотеки TCR согласно настоящему изобретению, при этом TCR сопоставляется с типом HLA образца.
В одном аспекте изобретение включает получение указания типа HLA индивидуума, у которого диагностирован NY-ESO-1/LAGE-1 положительный рак, выбор вектора экспрессии из библиотеки согласно настоящему изобретению на основе типа HLA и распределение вектора экспрессии для участия в применении при введении вектора экспрессии в иммунные клетки диагностированного индивидуума. Распределение вектора экспрессии может включать транспортирование вектора экспрессии с использованием любого приемлемого подхода.
В одном аспекте изобретение включает выбор вектора экспрессии из библиотеки согласно настоящему изобретению и введение вектора экспрессии в иммунные клетки, полученные от индивидуума, у которого диагностирован NY-ESO-/LAGE-11 положительный рак, причем тип HLA TCR, кодируемого вектором экспрессии, сопоставляется с типом HLA индивидуума. В вариантах осуществления изобретение, кроме того, включает введение иммунных клеток индивидууму, который нуждается в этом, который может представлять собой индивидуум, у которого был диагностирован NY-ESO-1/LAGE-1 положительный рак, или может представлять собой индивидуум с таким же типом HLA, как и у диагностированного индивидуума.
В одном аспекте изобретение включает исследование образца от индивидуума для того, чтобы определить, имеет ли или нет индивидуум NY-ESO-1/LAGE-1 положительный рак, и после определения того, что индивидуум имеет NY-ESO-1/LAGE-1 положительный рак, выбор вектора экспрессии из библиотеки согласно настоящему изобретению на основе типа HLA индивидуума, и введение вектора экспрессии в иммунные клетки индивидуума.
Следующие примеры предназначены для того, чтобы проиллюстрировать, а не ограничивать, изобретение.
Пример 1
В конкретных и иллюстративных вариантах осуществления полинуклеотидные последовательности, кодирующие TCR согласно изобретению, и аминокислотные последовательности α- и β-цепей TCR, кодированные полинуклеотидами, являются следующими. Типичные и неограничивающие примеры, демонстрирующие клонирование и применение TCR, представлены на Фигурах 1-4.

Рестрицируемый по HLA-A*02 NY-ESO-1157-165-специфический Т-клеточный клон "AL"
1. Нуклеотидная последовательность
α-цепь TCR

β-цепь TCR


2. Аминокислотная последовательность AL (начиная с инициаторного кодона, кодирующего метионин, до стоп-кодона, кодирующего терминацию (*))
α-цепь TCR

β-цепь TCR


Рестрицируемый по HLA-B*35NY-ESO-194-102-специфический Т-клеточный клон "KQ"
1. Нуклеотидная последовательность
α-цепь TCR

β-цепь TCR


2. Аминокислотная последовательность для KQ (начиная с инициаторного кодона, кодирующего метионин, до стоп-кодона, кодирующего терминацию (*))
α-цепь TCR

β-цепь TCR


Рестрицируемый по HLA-B*35 NY-ESO-194-104-специфический Т-клеточный клон "РР"
1. Нуклеотидная последовательность
α-цепь TCR

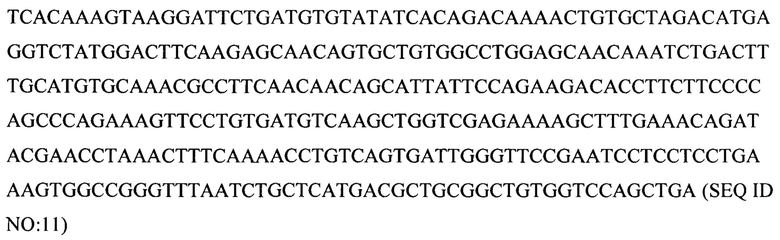
β-цепь TCR

2. Аминокислотная последовательность для РР (начиная с инициаторного кодона, кодирующего метионин, до стоп-кодона, кодирующего терминацию (*))
α-цепь TCR


β-цепь TCR


Рестрицируемый по HLA-B*27 NY-ESO-151-70-специфический Т-клеточный клон "19305CD8"
1. Нуклеотидная последовательность
α-цепь TCR

β-цепь TCR
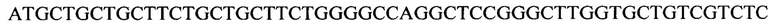

2. Аминокислотная последовательность для 19305CD8 (начиная с инициаторного кодона, кодирующего метионин, до стоп-кодона, кодирующего терминацию (*))
α-цепь TCR

β-цепь TCR


Рестрицируемый по HLA-Cw*15 NY-ESO-1127-135-специфический Т-клеточный клон "ВВ"
1. Нуклеотидная последовательность α-цепь TCR

β-цепь TCR


2. Аминокислотная последовательность для ВВ (начиная с инициаторного кодона, кодирующего метионин, до стоп-кодона, кодирующего терминацию (*))
α-цепь TCR

β-цепь TCR


Рестрицируемый по HLA-Cw*03 NY-ESO-192-100-специфический Т-клеточный клон "KB"
1. Нуклеотидная последовательность
α-цепь TCR


β-цепь TCR

2. Аминокислотная последовательность для KB (начиная с инициаторного кодона, кодирующего метионин, до стоп-кодона, кодирующего терминацию (*))
α-цепь TCR
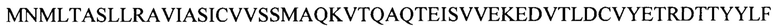

β-цепь TCR


Рестрицируемый по HLA-Cw*03 NY-ESO-196-104-специфический Т-клеточный клон "ST"
1. Нуклеотидная последовательность
α-цепь TCR

β-цепь TCR

2. Аминокислотная последовательность для ST (начиная с инициаторного кодона, кодирующего метионин, до стоп-кодона, кодирующего терминацию (*))
α-цепь TCR

β-цепь TCR



Рестрицируемый по HLA-A*02 NY-ESO-1157-165-специфический Т-клеточный клон "19305DP"
1. Нуклеотидная последовательность
α-цепь TCR

β-цепь TCR

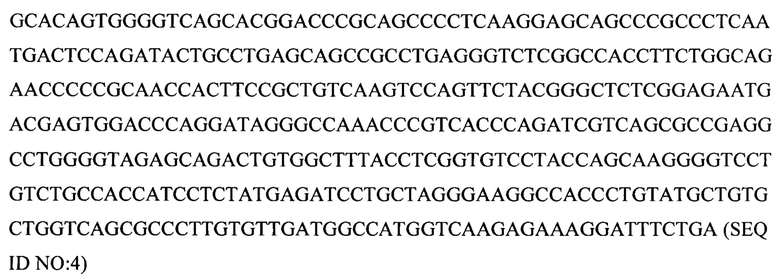
2. Аминокислотная последовательность для 19305DP (начиная с инициаторного кодона, кодирующего метионин, до стоп-кодона, кодирующего терминацию (*))
α-цепь TCR

β-цепь TCR


Рестрицируемый по HLA-A*02 NY-ESO-1157-165-специфический Т-клеточный клон "JD"
1. Нуклеотидная последовательность
α-цепь TCR


β-цепь TCR

2. Аминокислотная последовательность для JD (начиная с инициаторного кодона, кодирующего метионин до стоп-кодона, кодирующего терминацию (*))
α-цепь TCR

β-цепь TCR


Рестрицируемый по HLA-DRB1*07 NY-ESO-1(139-160)-специфический Т-клеточный клон "РВ-Р"
1. Нуклеотидная последовательность
α-цепь TCR

β-цепь TCR

2. Аминокислотная последовательность для РВ-Р (начиная с инициаторного кодона, кодирующего метионин, до стоп-кодона, кодирующего терминацию (*))
α-цепь TCR

β-цепь TCR


Рестрицируемый по HLA-DRJ3T*04 NY-ESO-1(111-143)-специфический Т-клеточный клон "РВ-Т"
1. Нуклеотидная последовательность
α-цепь TCR

β-цепь TCR


2. Аминокислотная последовательность для РВ-Т (начиная с инициаторного кодона, кодирующего метионин, до стоп-кодона, кодирующего терминацию (*))
α-цепь TCR

β-цепь TCR


Рестрицируемый по HLA-DRB1*07 HY-ESO-1(139-160)-специфический Т-клеточный клон "РВ13.2"
1. Нуклеотидная последовательность
α-цепь TCR

β-цепь TCR


2. Аминокислотная последовательность для РВ13.2 (начиная с инициаторного кодона, кодирующего метионин, до стоп-кодона, кодирующего терминацию (*))
α-цепь TCR

β-цепь TCR


Вместе с вышеприведенными последовательностями данного примера, этот пример раскрывает неограничивающую демонстрацию создания NY-ESO-1-специфичного TCR-экспрессирующего ретровирусного вектора (Фигура 1). Данные фигуры иллюстрируют следующее:
Фигура 1А. Для трансдукции Т-клеток, векторы, полученные из вирусов мышиных стволовых клеток (MSCV), широко использовались из-за сильной промоторной активности с помощью длинных концевых повторов (LTR) MSCV и стабильности in vivo трансгенной экспрессии в гемопоэтических клетках. Поскольку 3'-LTR копируется в 5'-LTR во время интеграции в клетки-хозяева, такие как Т-клетки, и является ответственным за транскрипцию трансгенов, 3'-LTR в плазмидах имеет важное значение в экспрессии. С другой стороны, 5'-LTR является ответственным за транскрипцию продуцирования вируса. Схематическое представление для классических векторов, полученных из MSCV (pMIG-II и pMIG-w), показано на Фигуре 1А. Оба вектора имеют LTR, полученный из MSCV, в 5' и 3' сайтах, сигнал упаковки множественные сайты клонирования (MCS). Трансген клонируют в MCS, за которым следует внутренний сайт связывания рибосомы (IRES) и ген зеленого флуоресцентного белка (GFP) для того, чтобы эффективно обнаруживать трансдуцированные клетки. Вектор pMIG-w имеет дополнительный посттранскрипционный регуляторный элемент вируса гепатита сурков (WRE), который усиливает экспрессию трансгена. В современные ретровирусные векторы вводятся дополнительные модификации, такие как те, которые обнаружены в коммерческом ретровирусном векторе, pDON-5 (Clontech). pDON-5, который получают из вектора вируса лейкоза мыши (MLV), заменяет 5'-LTR на CMV/MLV гибридный LTR для усиления продуцирования вируса посредством сильной промоторной активности CMV в клеточной линии, упаковывающей вирус. Кроме того, частичный интрон из гена фактора элонгации 1-альфа человека вводится для того, чтобы обеспечивать акцепторный сайт сплайсинга (SA), который вместе с эндогенным донорным сайтом сплайсинга (SD) индуцирует сплайсинг и усиливает экспрессию.
Фигура 1В. Для того чтобы создать ретровирусный вектор, который может продуцировать ретровирус с высоким титром, который индуцирует высокоуровневую трансгенную (TCR) экспрессию в Т-клетках, мы амплифицировали фрагмент ДНК из 5'-LTR в интрон, содержащий акцепторный сайт сплайсинга в pDON-5 плазмиде. Прямой праймер был сконструирован для добавления сайта распознавания рестрикционного фермента SgrAI перед 5'-LTR, и обратный праймер был сконструирован для добавления сайтов NotI и SalI после интрона. ПЦР-амплифицированный фрагмент обрабатывали SgrAI и SalI и вставляли в плазмиды pMIG-II и pMIG-w таким образом, чтобы заменить 5'-LTR на GFP.
Фигура 1С. Плазмиды, изображенные на Фигуре 1С, имеют только сайты распознавания NotI и SalI для клонирования. Использование SalI, которая распознает конкретную 6-мерную нуклеотидную последовательность, не является предпочтительным, поскольку часто может оказаться достаточным ее присутствия в трансгене, что, таким образом, в результате приводит к его расщеплению. Для обеспечения дополнительного сайта распознавания рестрикционного фермента, который, как полагают, вряд ли появятся у большинства трансгенов, мы амплифицировали фрагмент ДНК размером 1,8 тыс. осн. (лишний) с прямым праймером с сайтом рестрикции NotI, и обратным праймером с сайтами PacI-SalI. Амплифицированный фрагмент обрабатывали рестрикционными ферментами NotI и SalI и вставляли в новые плазмиды.
Фигура 1D. Изображенная кассета экспрессии TCR была амплифицирована прямым праймером с сайтом рестрикции NotI и обратным праймером с сайтом рестрикционного фермента PacI. Амплифицированную кассету экспрессии обрабатывали рестрикционными ферментами NotI и PacI и вставляли в новые плазмиды.
Трансдукция гена 19305DP-TCR в поликлонально активированные Т-клетки (Фигура 2). Т-клетки от здоровых индивидуумов были предварительно активированы в течение 2 дней с использованием фитогемаглютинина (РНА, полученного от Remel) в присутствии низкой дозы IL-2, IL-7 и IL-12. Ретровирусные частицы наносили на необработанный планшет для культур тканей, которые предварительно покрывали ретронектином (полученным от Clontech) и блокировали бычьим сывороточным альбумином (BSA от Sigma). Активированные Т-клетки инфицировали ретровирусом путем культивирования на планшете, покрытом ретровирусом. Вирусное инфицирование повторяли через 24 часа. Общее число инфицирования составляло 2. Инфицированные клетки размножали в присутствии IL-2 и IL-7. IL-12 включали в течение 5 дней после активации Т-клеток с РНА. После инфицирования, больше, чем 95% TCR ген-трансдуцированных Т-клеток экспрессировали трансдуцированные TCR путем окрашивания TCR Vβ подтип-специфическим антителом.
Противоопухолевая функция 19305DP-TCR-трансдуцированных Т-клеток (Фигуры 3 и 8-10). TCR ген-трансдуцированные Т-клетки совместно культивировали с HLA-A*02+NY-ESO-1+ меланомной клеточной линией, SK-MEL-37 или HLA-A*02+NY-ESO-1- меланомной клеточной линией, SK-MEL-29 в течение 6 часов в присутствии Golgi Stop (BD Biosciences). Клетки окрашивали анти-CD4 и анти-CD8 антителами, конъюгированными с флуорохромом, и фиксировали, и пермеабилизировали с использованием набора BD Cytofix/Cytoperm (BD Biosciences). Внутриклеточное IFN-γ окрашивали анти-IFN-γ антителом, конъюгированным с флуорохромом. Обе TCR ген-трансдуцированные CD4+ и CD8+ Т-клетки продуцировали IFN-γ только тогда, когда они были совместно культивированы с NY-ESO-1+SK-MEL-37, а не с SK-MEL-29. Нетрансдуцированные CD4+ и CD8+ Т-клетки не продуцировали IFN-γ против SK-MEL-37 или SK-MEL-29 (Фигура 3). На Фигуре 8, иммунодефицитным NOD/SCID/IL-2Rγ-цепь-дефицитным (NSG) мышам инокулировали 1 миллион SK-MEL-37. После того, как рост опухоли был подтвержден на 9-й день, инъекционно вводили 10 миллионов 19305DP-TCR ген-трансдуцированных Т-клеток. Контрольным мышам с опухолью инъекционно вводили нерелевантные TCR-трансдуцированные Т-клетки. Мыши, которым вводили 19305DP-TCR-трансдуцированные Т-клетки, показали значительную задержку роста опухоли, и большинство мышей в конечном итоге отторгали опухолевый ксенотрансплантат. Опухоль непрерывно росла у контрольных мышей, получавших нерелевантные TCR-трансдуцированные Т-клетки.
Пример 2
Данный пример приводит описание дополнительных TCR последовательностей, которые могут быть включены в любую из TCR последовательностей, описанных в Примере 1 в библиотеках согласно настоящему изобретению. Данные TCR наделяют CD4+ Т-клетки способностью непосредственно распознавать NY-ESO-1/LAGE-1 положительные раковые клетки.
"JM" Рестрицируемый по HLA-DPB1*0401/0402 NY-ESO-1157-170-специфический распознающий опухоль CD4+ Т-клеточный клон
(а) нуклеотидные последовательности кДНК α- и β-цепей TCR
α-цепь TCR


β-цепь TCR

(b) аминокислотные последовательности α- и β-цепей TCR для JM (вариабельные области TCR выделены курсивом, области CDR3 выделены жирным шрифтом),
α-цепь TCR

β-цепь TCR

"5В8" Рестрицируемый по HLA-DPB1*0401/0402 NY-ESO-1157-170-специфический распознающий опухоль CD4+ Т-клеточный клон
(а) нуклеотидные последовательности кДНК α- и β-цепей TCR
α-цепь TCR

β-цепь TCR


(b) аминокислотные последовательности α- и β-цепей TCR для 5В8 (вариабельные области TCR выделены курсивом, области CDR3 выделены жирным шрифтом)
α-цепь TCR

β-цепь TCR


"SB95" Рестрицируемый по HLA-DRB1*0101 NY-ESO-195-106-специфический распознающий опухоль CD4+ Т-клеточный клон
(а) нуклеотидные последовательности кДНК α- и β-цепей TCR
α-цепь TCR

β-цепь TCR

(b) аминокислотные последовательности α- и β-цепей TCR для SB95 (вариабельные области TCR выделены курсивом, области CDR3 выделены жирным шрифтом)
α-цепь TCR

β-цепь TCR

На Фигуре 4 приведен неограничивающий пример вектора экспрессии, который может быть использован для экспрессии любого из TCR из данного Примера. Для Фигуры 4: (А) Ретровирусный вектор, использованный для экспрессии TCR. LTR: длинный концевой повтор; ψ: сигнал упаковки; MCS: множественный сайт клонирования; IRES: внутренний сайт посадки рибосомы; eGFP: усиленный зеленый флуоресцентный белок. (В) TCR экспрессирующая кассета. (I) последовательности кДНК, кодирующие β- и α-цепи TCR, соединяются линкером GSG (Gly-Ser-Gly) и последовательностью Р2А рибосомального пропуска. (II) последовательности кДНК, кодирующие β- и α-цепи TCR, соединяются через сайт распознавания фуринпротеазы (RAKR (Arg-Ala-Lys-Arg), линкер SGSG (Ser-Gly-Ser-Gly), эпитоп V5 и последовательность Р2А рибосомального пропуска.
Как описано в РСТ PCT/US14/25673, TCR из данного Примера способны стимулировать непосредственное распознавание раковых клеток и индуцировать их апоптоз. Кроме того, CD4+ Т-клетки, экспрессирующие TCR из данного Примера, как было обнаружено, эффективно увеличивают цитотоксическую активность опухолево-антигенспецифических CD8+ Т-клеток за счет непосредственного распознавания раковых клеток в отсутствие антиген-представляющих клеток. Кроме того, CD8+ Т-клетки, совместно стимулированные с CD4+ Т-клетками, экспрессирующими рекомбинантные TCR из данного Примера, активно пролиферировали и повышающе регулировали маркеры центральных Т-клеток памяти, a CD4+ Т-клетки, экспрессирующие данные TCR, показали значительную противоопухолевую активность in vivo для того, чтобы ингибировать рост раковых клеток человека у иммунодефицитных мышей, и данные CD4+ Т-клетки с опухолево-антигенспецифическими CD8+ Т-клетками совместно ингибировали рост опухоли in vivo.
Аллельная частота HLA для разных этнических групп в Соединенных Штатах. (Показана для 10 наиболее часто встречающихся типов у американцев европейского происхождения в Таблице 1). Данные для HLA-A,B,C,DR и DQ были получены и модифицированы от The National Marrow Donor Program®(NMDP)/Be The Match®, вебсайт: bioinformatics.bethematchclinical.org/. Данные для HLA-DP были получены от The Allele Frequency Net Database: www.allelefrequencies.net/contact.asp.
В варианте осуществления, библиотека согласно настоящему изобретению содержит множество NY-ESO-1-специфичных TCR, рестрицируемых по типам HLA в Таблице 1 ниже, выделенным подчеркиванием/жирным шрифтом.

Хотя изобретение было в частности показано и описано со ссылкой на конкретные варианты осуществления, специалистам в данной области техники должно быть понятно, что в него могут быть внесены различные изменения по форме и в деталях, не отступая от сущности и объема настоящего изобретения, как раскрыто в данной заявке.
0069.0007RU1 WO/2017/120428
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
SEQUENCE LISTING
<110> Health Research, Inc.
<120> COMPOSITIONS AND LIBRARIES COMPRISING RECOMBINANT T-CELL
RECEPTORS AND METHODS OF USING RECOMBINANT T-CELL RECEPTORS
<130> 003551.00681
<140> US 62/275,600
<141> 2016-01-06
<160> 66
<170> PatentIn version 3.5
<210> 1
<211> 270
<212> PRT
<213> Human
<400> 1
Met Glu Thr Leu Leu Gly Val Ser Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ala Arg Val Asn Ser Gln Gln Gly Glu Glu Asp Pro Gln Ala Leu Ser
20 25 30
Ile Gln Glu Gly Glu Asn Ala Thr Met Asn Cys Ser Tyr Lys Thr Ser
35 40 45
Ile Asn Asn Leu Gln Trp Tyr Arg Gln Asn Ser Gly Arg Gly Leu Val
50 55 60
His Leu Ile Leu Ile Arg Ser Asn Glu Arg Glu Lys His Ser Gly Arg
65 70 75 80
Leu Arg Val Thr Leu Asp Thr Ser Lys Lys Ser Ser Ser Leu Leu Ile
85 90 95
Thr Ala Ser Arg Ala Ala Asp Thr Ala Ser Tyr Phe Cys Ala Thr Asp
100 105 110
Gly Gly Gly Thr Leu Thr Phe Gly Lys Gly Thr Met Leu Leu Val Ser
115 120 125
Pro Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
130 135 140
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
145 150 155 160
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
165 170 175
Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
180 185 190
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
195 200 205
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
210 215 220
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
225 230 235 240
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
245 250 255
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265 270
<210> 2
<211> 308
<212> PRT
<213> Human
<400> 2
Met Asp Ser Trp Thr Leu Cys Cys Val Ser Leu Cys Ile Leu Val Ala
1 5 10 15
Lys His Thr Asp Ala Gly Val Ile Gln Ser Pro Arg His Glu Val Thr
20 25 30
Glu Met Gly Gln Glu Val Thr Leu Arg Cys Lys Pro Ile Ser Gly His
35 40 45
Asp Tyr Leu Phe Trp Tyr Arg Gln Thr Met Met Arg Gly Leu Glu Leu
50 55 60
Leu Ile Tyr Phe Asn Asn Asn Val Pro Ile Asp Asp Ser Gly Met Pro
65 70 75 80
Glu Asp Arg Phe Ser Ala Lys Met Pro Asn Ala Ser Phe Ser Thr Leu
85 90 95
Lys Ile Gln Pro Ser Glu Pro Arg Asp Ser Ala Val Tyr Phe Cys Ala
100 105 110
Ser Lys Trp Gly Gly Thr Glu Ala Phe Phe Gly Gln Gly Thr Arg Leu
115 120 125
Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val
130 135 140
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
145 150 155 160
Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp
165 170 175
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln
180 185 190
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
195 200 205
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
210 215 220
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
225 230 235 240
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
245 250 255
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly
260 265 270
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
275 280 285
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
290 295 300
Arg Lys Asp Phe
305
<210> 3
<211> 813
<212> DNA
<213> Human
<400> 3
atggaaactc tcctgggagt gtctttggtg attctatggc ttcaactggc tagggtgaac 60
agtcaacagg gagaagagga tcctcaggcc ttgagcatcc aggagggtga aaatgccacc 120
atgaactgca gttacaaaac tagtataaac aatttacagt ggtatagaca aaattcaggt 180
agaggccttg tccacctaat tttaatacgt tcaaatgaaa gagagaaaca cagtggaaga 240
ttaagagtca cgcttgacac ttccaagaaa agcagttcct tgttgatcac ggcttcccgg 300
gcagcagaca ctgcttctta cttctgtgct acggacgggg ggggcaccct cacctttggg 360
aaggggacta tgcttctagt ctctccagat atccagaacc ctgaccctgc cgtgtaccag 420
ctgagagact ctaaatccag tgacaagtct gtctgcctat tcaccgattt tgattctcaa 480
acaaatgtgt cacaaagtaa ggattctgat gtgtatatca cagacaaaac tgtgctagac 540
atgaggtcta tggacttcaa gagcaacagt gctgtggcct ggagcaacaa atctgacttt 600
gcatgtgcaa acgccttcaa caacagcatt attccagaag acaccttctt ccccagccca 660
gaaagttcct gtgatgtcaa gctggtcgag aaaagctttg aaacagatac gaacctaaac 720
tttcaaaacc tgtcagtgat tgggttccga atcctcctcc tgaaagtggc cgggtttaat 780
ctgctcatga cgctgcggct gtggtccagc tga 813
<210> 4
<211> 927
<212> DNA
<213> Human
<400> 4
atggactcct ggaccctctg ctgtgtgtcc ctttgcatcc tggtagcaaa gcacacagat 60
gctggagtta tccagtcacc ccggcacgag gtgacagaga tgggacaaga agtgactctg 120
agatgtaaac caatttcagg acacgactac cttttctggt acagacagac catgatgcgg 180
ggactggagt tgctcattta ctttaacaac aacgttccga tagatgattc agggatgccc 240
gaggatcgat tctcagctaa gatgcctaat gcatcattct ccactctgaa gatccagccc 300
tcagaaccca gggactcagc tgtgtacttc tgtgccagca agtggggcgg cactgaagct 360
ttctttggac aaggcaccag actcacagtt gtagaggacc tgaacaaggt gttcccaccc 420
gaggtcgctg tgtttgagcc atcagaagca gagatctccc acacccaaaa ggccacactg 480
gtgtgcctgg ccacaggctt cttccctgac cacgtggagc tgagctggtg ggtgaatggg 540
aaggaggtgc acagtggggt cagcacggac ccgcagcccc tcaaggagca gcccgccctc 600
aatgactcca gatactgcct gagcagccgc ctgagggtct cggccacctt ctggcagaac 660
ccccgcaacc acttccgctg tcaagtccag ttctacgggc tctcggagaa tgacgagtgg 720
acccaggata gggccaaacc cgtcacccag atcgtcagcg ccgaggcctg gggtagagca 780
gactgtggct ttacctcggt gtcctaccag caaggggtcc tgtctgccac catcctctat 840
gagatcctgc tagggaaggc caccctgtat gctgtgctgg tcagcgccct tgtgttgatg 900
gccatggtca agagaaagga tttctga 927
<210> 5
<211> 277
<212> PRT
<213> Human
<400> 5
Met Met Lys Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ser Trp Val Trp Ser Gln Gln Lys Glu Val Glu Gln Asn Ser Gly Pro
20 25 30
Leu Ser Val Pro Glu Gly Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser
35 40 45
Asp Arg Gly Ser Gln Ser Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys
50 55 60
Ser Pro Glu Leu Ile Met Ser Ile Tyr Ser Asn Gly Asp Lys Glu Asp
65 70 75 80
Gly Arg Phe Thr Ala Gln Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu
85 90 95
Leu Ile Arg Asp Ser Gln Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala
100 105 110
Val Gly Gly Leu Thr Ser Ser Asn Thr Gly Lys Leu Ile Phe Gly Gln
115 120 125
Gly Thr Thr Leu Gln Val Lys Pro Asp Ile Gln Asn Pro Asp Pro Ala
130 135 140
Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu
145 150 155 160
Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser
165 170 175
Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp
180 185 190
Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala
195 200 205
Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe
210 215 220
Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe
225 230 235 240
Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe
245 250 255
Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu
260 265 270
Arg Leu Trp Ser Ser
275
<210> 6
<211> 307
<212> PRT
<213> Human
<400> 6
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Asn Gln Ile Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg Leu Thr
115 120 125
Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe
130 135 140
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
145 150 155 160
Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp
165 170 175
Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro
180 185 190
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
195 200 205
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
210 215 220
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
225 230 235 240
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
245 250 255
Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val
260 265 270
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
275 280 285
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
290 295 300
Lys Asp Phe
305
<210> 7
<211> 834
<212> DNA
<213> Human
<400> 7
atgatggcag gcattcgagc tttatttatg tacttgtggc tgcagctgga ctgggtgagc 60
agaggagaga gtgtggggct gcatcttcct accctgagtg tccaggaggg tgacaactct 120
attatcaact gtgcttattc aaacagcgcc tcagactact tcatttggta caagcaagaa 180
tctggaaaag gtcctcaatt cattatagac attcgttcaa atatggacaa aaggcaaggc 240
caaagagtca ccgttttatt gaataagaca gtgaaacatc tctctctgca aattgcagct 300
actcaacctg gagactcagc tgtctacttt tgtgcagaga ataccgcccc acataatgca 360
ggcaacatgc tcacctttgg agggggaaca aggttaatgg tcaaacccca tatccagaac 420
cctgaccctg ccgtgtacca gctgagagac tctaaatcca gtgacaagtc tgtctgccta 480
ttcaccgatt ttgattctca aacaaatgtg tcacaaagta aggattctga tgtgtatatc 540
acagacaaaa ctgtgctaga catgaggtct atggacttca agagcaacag tgctgtggcc 600
tggagcaaca aatctgactt tgcatgtgca aacgccttca acaacagcat tattccagaa 660
gacaccttct tccccagccc agaaagttcc tgtgatgtca agctggtcga gaaaagcttt 720
gaaacagata cgaacctaaa ctttcaaaac ctgtcagtga ttgggttccg aatcctcctc 780
ctgaaagtgg ccgggtttaa tctgctcatg acgctgcggc tgtggtccag ctga 834
<210> 8
<211> 933
<212> DNA
<213> Human
<400> 8
atgggccctg ggctcctctg ctgggtgctg ctttgtctcc tgggagcagg cccagtggac 60
gctggagtca cccaaagtcc cacacacctg atcaaaacga gaggacagca agtgactctg 120
agatgctctc ctatctctgg gcacaagagt gtgtcctggt accaacaggt cctgggtcag 180
gggccccagt ttatctttca gtattatgag aaagaagaga gaggaagagg aaacttccct 240
gatcgattct cagctcgcca gttccctaac tatagctctg agctgaatgt gaacgccttg 300
ttgctggggg actcggccct gtatctctgt gccagcagct acgacagggg gataaactat 360
ggctacacct tcggttcggg gaccaggtta accgttgtag aggacctgaa caaggtgttc 420
ccacccgagg tcgctgtgtt tgagccatca gaagcagaga tctcccacac ccaaaaggcc 480
acactggtgt gcctggccac aggcttcttc cctgaccacg tggagctgag ctggtgggtg 540
aatgggaagg aggtgcacag tggggtcagc acggacccgc agcccctcaa ggagcagccc 600
gccctcaatg actccagata ctgcctgagc agccgcctga gggtctcggc caccttctgg 660
cagaaccccc gcaaccactt ccgctgtcaa gtccagttct acgggctctc ggagaatgac 720
gagtggaccc aggatagggc caaacccgtc acccagatcg tcagcgccga ggcctggggt 780
agagcagact gtggctttac ctcggtgtcc taccagcaag gggtcctgtc tgccaccatc 840
ctctatgaga tcctgctagg gaaggccacc ctgtatgctg tgctggtcag cgcccttgtg 900
ttgatggcca tggtcaagag aaaggatttc tga 933
<210> 9
<211> 277
<212> PRT
<213> Human
<400> 9
Met Met Ala Gly Ile Arg Ala Leu Phe Met Tyr Leu Trp Leu Gln Leu
1 5 10 15
Asp Trp Val Ser Arg Gly Glu Ser Val Gly Leu His Leu Pro Thr Leu
20 25 30
Ser Val Gln Glu Gly Asp Asn Ser Ile Ile Asn Cys Ala Tyr Ser Asn
35 40 45
Ser Ala Ser Asp Tyr Phe Ile Trp Tyr Lys Gln Glu Ser Gly Lys Gly
50 55 60
Pro Gln Phe Ile Ile Asp Ile Arg Ser Asn Met Asp Lys Arg Gln Gly
65 70 75 80
Gln Arg Val Thr Val Leu Leu Asn Lys Thr Val Lys His Leu Ser Leu
85 90 95
Gln Ile Ala Ala Thr Gln Pro Gly Asp Ser Ala Val Tyr Phe Cys Ala
100 105 110
Glu Asn Thr Ala Pro His Asn Ala Gly Asn Met Leu Thr Phe Gly Gly
115 120 125
Gly Thr Arg Leu Met Val Lys Pro His Ile Gln Asn Pro Asp Pro Ala
130 135 140
Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu
145 150 155 160
Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser
165 170 175
Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp
180 185 190
Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala
195 200 205
Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe
210 215 220
Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe
225 230 235 240
Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe
245 250 255
Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu
260 265 270
Arg Leu Trp Ser Ser
275
<210> 10
<211> 310
<212> PRT
<213> Human
<400> 10
Met Gly Pro Gly Leu Leu Cys Trp Val Leu Leu Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Asp Ala Gly Val Thr Gln Ser Pro Thr His Leu Ile Lys
20 25 30
Thr Arg Gly Gln Gln Val Thr Leu Arg Cys Ser Pro Ile Ser Gly His
35 40 45
Lys Ser Val Ser Trp Tyr Gln Gln Val Leu Gly Gln Gly Pro Gln Phe
50 55 60
Ile Phe Gln Tyr Tyr Glu Lys Glu Glu Arg Gly Arg Gly Asn Phe Pro
65 70 75 80
Asp Arg Phe Ser Ala Arg Gln Phe Pro Asn Tyr Ser Ser Glu Leu Asn
85 90 95
Val Asn Ala Leu Leu Leu Gly Asp Ser Ala Leu Tyr Leu Cys Ala Ser
100 105 110
Ser Tyr Asp Arg Gly Ile Asn Tyr Gly Tyr Thr Phe Gly Ser Gly Thr
115 120 125
Arg Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe
305 310
<210> 11
<211> 822
<212> DNA
<213> Human
<400> 11
atggcctctg cacccatctc gatgcttgcg atgctcttca cattgagtgg gctgagagct 60
cagtcagtgg ctcagccgga agatcaggtc aacgttgctg aagggaatcc tctgactgtg 120
aaatgcacct attcagtctc tggaaaccct tatctttttt ggtatgttca ataccccaac 180
cgaggcctcc agttccttct gaaatacatc acaggggata acctggttaa aggcagctat 240
ggctttgaag ctgaatttaa caagagccaa acctccttcc acctgaagaa accatctgcc 300
cttgtgagcg actccgcttt gtacttctgt gctgtgagag atgttgtgga ggggaaattg 360
cagtttggag cagggaccca ggttgtggtc accccagata tccagaaccc tgaccctgcc 420
gtgtaccagc tgagagactc taaatccagt gacaagtctg tctgcctatt caccgatttt 480
gattctcaaa caaatgtgtc acaaagtaag gattctgatg tgtatatcac agacaaaact 540
gtgctagaca tgaggtctat ggacttcaag agcaacagtg ctgtggcctg gagcaacaaa 600
tctgactttg catgtgcaaa cgccttcaac aacagcatta ttccagaaga caccttcttc 660
cccagcccag aaagttcctg tgatgtcaag ctggtcgaga aaagctttga aacagatacg 720
aacctaaact ttcaaaacct gtcagtgatt gggttccgaa tcctcctcct gaaagtggcc 780
gggtttaatc tgctcatgac gctgcggctg tggtccagct ga 822
<210> 12
<211> 936
<212> DNA
<213> Human
<400> 12
atgggcacca gcctcctctg ctggatggcc ctgtgtctcc tgggggcaga tcacgcagat 60
actggagtct cccaggaccc cagacacaag atcacaaaga ggggacagaa tgtaactttc 120
aggtgtgatc caatttctga acacaaccgc ctttattggt accgacagac cctggggcag 180
ggcccagagt ttctgactta cttccagaat gaagctcaac tagaaaaatc aaggctgctc 240
agtgatcggt tctctgcaga gaggcctaag ggatctttct ccaccttgga gatccagcgc 300
acagagcagg gggactcggc catgtatctc tgtgccagca gcaccacaag ctcctacgag 360
cagtacttcg ggccgggcac caggctcacg gtcacagagg acctgaaaaa cgtgttccca 420
cccgaggtcg ctgtgtttga gccatcagaa gcagagatct cccacaccca aaaggccaca 480
ctggtgtgcc tggccacagg cttctacccc gaccacgtgg agctgagctg gtgggtgaat 540
gggaaggagg tgcacagtgg ggtcagcaca gacccgcagc ccctcaagga gcagcccgcc 600
ctcaatgact ccagatactg cctgagcagc cgcctgaggg tctcggccac cttctggcag 660
aacccccgca accacttccg ctgtcaagtc cagttctacg ggctctcgga gaatgacgag 720
tggacccagg atagggccaa acctgtcacc cagatcgtca gcgccgaggc ctggggtaga 780
gcagactgtg gcttcacctc cgagtcttac cagcaagggg tcctgtctgc caccatcctc 840
tatgagatct tgctagggaa ggccaccttg tatgccgtgc tggtcagtgc cctcgtgctg 900
atggccatgg tcaagagaaa ggattccaga ggctag 936
<210> 13
<211> 273
<212> PRT
<213> Human
<400> 13
Met Ala Ser Ala Pro Ile Ser Met Leu Ala Met Leu Phe Thr Leu Ser
1 5 10 15
Gly Leu Arg Ala Gln Ser Val Ala Gln Pro Glu Asp Gln Val Asn Val
20 25 30
Ala Glu Gly Asn Pro Leu Thr Val Lys Cys Thr Tyr Ser Val Ser Gly
35 40 45
Asn Pro Tyr Leu Phe Trp Tyr Val Gln Tyr Pro Asn Arg Gly Leu Gln
50 55 60
Phe Leu Leu Lys Tyr Ile Thr Gly Asp Asn Leu Val Lys Gly Ser Tyr
65 70 75 80
Gly Phe Glu Ala Glu Phe Asn Lys Ser Gln Thr Ser Phe His Leu Lys
85 90 95
Lys Pro Ser Ala Leu Val Ser Asp Ser Ala Leu Tyr Phe Cys Ala Val
100 105 110
Arg Asp Val Val Glu Gly Lys Leu Gln Phe Gly Ala Gly Thr Gln Val
115 120 125
Val Val Thr Pro Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
130 135 140
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
145 150 155 160
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
165 170 175
Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
180 185 190
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
195 200 205
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
210 215 220
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
225 230 235 240
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
245 250 255
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
260 265 270
Ser
<210> 14
<211> 311
<212> PRT
<213> Human
<400> 14
Met Gly Thr Ser Leu Leu Cys Trp Met Ala Leu Cys Leu Leu Gly Ala
1 5 10 15
Asp His Ala Asp Thr Gly Val Ser Gln Asp Pro Arg His Lys Ile Thr
20 25 30
Lys Arg Gly Gln Asn Val Thr Phe Arg Cys Asp Pro Ile Ser Glu His
35 40 45
Asn Arg Leu Tyr Trp Tyr Arg Gln Thr Leu Gly Gln Gly Pro Glu Phe
50 55 60
Leu Thr Tyr Phe Gln Asn Glu Ala Gln Leu Glu Lys Ser Arg Leu Leu
65 70 75 80
Ser Asp Arg Phe Ser Ala Glu Arg Pro Lys Gly Ser Phe Ser Thr Leu
85 90 95
Glu Ile Gln Arg Thr Glu Gln Gly Asp Ser Ala Met Tyr Leu Cys Ala
100 105 110
Ser Ser Thr Thr Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 15
<211> 825
<212> DNA
<213> Human
<400> 15
atgatggcag gcattcgagc tttatttatg tacttgtggc tgcagctgga ctgggtgagc 60
agaggagaga gtgtggggct gcatcttcct accctgagtg tccaggaggg tgacaactct 120
attatcaact gtgcttattc aaacagcgcc tcagactact tcatttggta caagcaagaa 180
tctggaaaag gtcctcaatt cattatagac attcgttcaa atatggacaa aaggcaaggc 240
caaagagtca ccgttttatt gaataagaca gtgaaacatc tctctctgca aattgcagct 300
actcaacctg gagactcagc tgtctacttt tgtgcagagg cgggaggaag ccaaggaaat 360
ctcatctttg gaaaaggcac taaactctct gttaaaccaa atatccagaa ccctgaccct 420
gccgtgtacc agctgagaga ctctaaatcc agtgacaagt ctgtctgcct attcaccgat 480
tttgattctc aaacaaatgt gtcacaaagt aaggattctg atgtgtatat cacagacaaa 540
actgtgctag acatgaggtc tatggacttc aagagcaaca gtgctgtggc ctggagcaac 600
aaatctgact ttgcatgtgc aaacgccttc aacaacagca ttattccaga agacaccttc 660
ttccccagcc cagaaagttc ctgtgatgtc aagctggtcg agaaaagctt tgaaacagat 720
acgaacctaa actttcaaaa cctgtcagtg attgggttcc gaatcctcct cctgaaagtg 780
gccgggttta atctgctcat gacgctgcgg ctgtggtcca gctga 825
<210> 16
<211> 942
<212> DNA
<213> Human
<400> 16
atgggccccg ggctcctctg ctgggcactg ctttgtctcc tgggagcagg cttagtggac 60
gctggagtca cccaaagtcc cacacacctg atcaaaacga gaggacagca agtgactctg 120
agatgctctc ctaagtctgg gcatgacact gtgtcctggt accaacaggc cctgggtcag 180
gggccccagt ttatctttca gtattatgag gaggaagaga gacagagagg caacttccct 240
gatcgattct caggtcacca gttccctaac tatagctctg agctgaatgt gaacgccttg 300
ttgctggggg actcggccct ctatctctgt gccagcagct tttggggtcg ttctcaccct 360
tcaaactatg gctacacctt cggttcgggg accaggttaa ccgttgtaga ggacctgaac 420
aaggtgttcc cacccgaggt cgctgtgttt gagccatcag aagcagagat ctcccacacc 480
caaaaggcca cactggtgtg cctggccaca ggcttcttcc ctgaccacgt ggagctgagc 540
tggtgggtga atgggaagga ggtgcacagt ggggtcagca cggacccgca gcccctcaag 600
gagcagcccg ccctcaatga ctccagatac tgcctgagca gccgcctgag ggtctcggcc 660
accttctggc agaacccccg caaccacttc cgctgtcaag tccagttcta cgggctctcg 720
gagaatgacg agtggaccca ggatagggcc aaacccgtca cccagatcgt cagcgccgag 780
gcctggggta gagcagactg tggctttacc tcggtgtcct accagcaagg ggtcctgtct 840
gccaccatcc tctatgagat cctgctaggg aaggccaccc tgtatgctgt gctggtcagc 900
gcccttgtgt tgatggccat ggtcaagaga aaggatttct ga 942
<210> 17
<211> 274
<212> PRT
<213> Human
<400> 17
Met Met Ala Gly Ile Arg Ala Leu Phe Met Tyr Leu Trp Leu Gln Leu
1 5 10 15
Asp Trp Val Ser Arg Gly Glu Ser Val Gly Leu His Leu Pro Thr Leu
20 25 30
Ser Val Gln Glu Gly Asp Asn Ser Ile Ile Asn Cys Ala Tyr Ser Asn
35 40 45
Ser Ala Ser Asp Tyr Phe Ile Trp Tyr Lys Gln Glu Ser Gly Lys Gly
50 55 60
Pro Gln Phe Ile Ile Asp Ile Arg Ser Asn Met Asp Lys Arg Gln Gly
65 70 75 80
Gln Arg Val Thr Val Leu Leu Asn Lys Thr Val Lys His Leu Ser Leu
85 90 95
Gln Ile Ala Ala Thr Gln Pro Gly Asp Ser Ala Val Tyr Phe Cys Ala
100 105 110
Glu Ala Gly Gly Ser Gln Gly Asn Leu Ile Phe Gly Lys Gly Thr Lys
115 120 125
Leu Ser Val Lys Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
130 135 140
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
145 150 155 160
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
165 170 175
Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
180 185 190
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
195 200 205
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
210 215 220
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
225 230 235 240
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
245 250 255
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
260 265 270
Ser Ser
<210> 18
<211> 313
<212> PRT
<213> Human
<400> 18
Met Gly Pro Gly Leu Leu Cys Trp Ala Leu Leu Cys Leu Leu Gly Ala
1 5 10 15
Gly Leu Val Asp Ala Gly Val Thr Gln Ser Pro Thr His Leu Ile Lys
20 25 30
Thr Arg Gly Gln Gln Val Thr Leu Arg Cys Ser Pro Lys Ser Gly His
35 40 45
Asp Thr Val Ser Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Gln Phe
50 55 60
Ile Phe Gln Tyr Tyr Glu Glu Glu Glu Arg Gln Arg Gly Asn Phe Pro
65 70 75 80
Asp Arg Phe Ser Gly His Gln Phe Pro Asn Tyr Ser Ser Glu Leu Asn
85 90 95
Val Asn Ala Leu Leu Leu Gly Asp Ser Ala Leu Tyr Leu Cys Ala Ser
100 105 110
Ser Phe Trp Gly Arg Ser His Pro Ser Asn Tyr Gly Tyr Thr Phe Gly
115 120 125
Ser Gly Thr Arg Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro
130 135 140
Pro Glu Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr
145 150 155 160
Gln Lys Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His
165 170 175
Val Glu Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val
180 185 190
Ser Thr Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser
195 200 205
Arg Tyr Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln
210 215 220
Asn Pro Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser
225 230 235 240
Glu Asn Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile
245 250 255
Val Ser Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val
260 265 270
Ser Tyr Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu
275 280 285
Leu Gly Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu
290 295 300
Met Ala Met Val Lys Arg Lys Asp Phe
305 310
<210> 19
<211> 840
<212> DNA
<213> Human
<400> 19
atgaacatgc tgactgccag cctgttgagg gcagtcatag cctccatctg tgttgtatcc 60
agcatggctc agaaggtaac tcaagcgcag actgaaattt ctgtggtgga gaaggaggat 120
gtgaccttgg actgtgtgta tgaaacccgt gatactactt attacttatt ctggtacaag 180
caaccaccaa gtggagaatt ggttttcctt attcgtcgga actcttttga tgagcaaaat 240
gaaataagtg gtcggtattc ttggaacttc cagaaatcca ccagttcctt caacttcacc 300
atcacagcct cacaagtcgt ggactcagca gtatacttct gtgctctgag tgaggcaagc 360
gggagagatg acaagatcat ctttggaaaa gggacacgac ttcatattct ccccaatatc 420
cagaaccctg accctgccgt gtaccagctg agagactcta aatccagtga caagtctgtc 480
tgcctattca ccgattttga ttctcaaaca aatgtgtcac aaagtaagga ttctgatgtg 540
tatatcacag acaaaactgt gctagacatg aggtctatgg acttcaagag caacagtgct 600
gtggcctgga gcaacaaatc tgactttgca tgtgcaaacg ccttcaacaa cagcattatt 660
ccagaagaca ccttcttccc cagcccagaa agttcctgtg atgtcaagct ggtcgagaaa 720
agctttgaaa cagatacgaa cctaaacttt caaaacctgt cagtgattgg gttccgaatc 780
ctcctcctga aagtggccgg gtttaatctg ctcatgacgc tgcggctgtg gtccagctga 840
<210> 20
<211> 930
<212> DNA
<213> Human
<400> 20
atgctgctgc ttctgctgct tctggggcca ggctccgggc ttggtgctgt cgtctctcaa 60
catccgagct gggttatctg taagagtgga acctctgtga agatcgagtg ccgttccctg 120
gactttcagg ccacaactat gttttggtat cgtcagttcc cgaaacagag tctcatgctg 180
atggcaactt ccaatgaggg ctccaaggcc acatacgagc aaggcgtcga gaaggacaag 240
tttctcatca accatgcaag cctgaccttg tccactctga cagtgaccag tgcccatcct 300
gaagacagca gcttctacat ctgcagtgct agagtcgact ttgaccgtga cgagcagttc 360
ttcgggccag ggacacggct caccgtgcta gaggacctga aaaacgtgtt cccacccgag 420
gtcgctgtgt ttgagccatc agaagcagag atctcccaca cccaaaaggc cacactggtg 480
tgcctggcca caggcttcta ccccgaccac gtggagctga gctggtgggt gaatgggaag 540
gaggtgcaca gtggggtcag cacagacccg cagcccctca aggagcagcc cgccctcaat 600
gactccagat actgcctgag cagccgcctg agggtctcgg ccaccttctg gcagaacccc 660
cgcaaccact tccgctgtca agtccagttc tacgggctct cggagaatga cgagtggacc 720
caggataggg ccaaacctgt cacccagatc gtcagcgccg aggcctgggg tagagcagac 780
tgtggcttca cctccgagtc ttaccagcaa ggggtcctgt ctgccaccat cctctatgag 840
atcttgctag ggaaggccac cttgtatgcc gtgctggtca gtgccctcgt gctgatggcc 900
atggtcaaga gaaaggattc cagaggctag 930
<210> 21
<211> 279
<212> PRT
<213> Human
<400> 21
Met Asn Met Leu Thr Ala Ser Leu Leu Arg Ala Val Ile Ala Ser Ile
1 5 10 15
Cys Val Val Ser Ser Met Ala Gln Lys Val Thr Gln Ala Gln Thr Glu
20 25 30
Ile Ser Val Val Glu Lys Glu Asp Val Thr Leu Asp Cys Val Tyr Glu
35 40 45
Thr Arg Asp Thr Thr Tyr Tyr Leu Phe Trp Tyr Lys Gln Pro Pro Ser
50 55 60
Gly Glu Leu Val Phe Leu Ile Arg Arg Asn Ser Phe Asp Glu Gln Asn
65 70 75 80
Glu Ile Ser Gly Arg Tyr Ser Trp Asn Phe Gln Lys Ser Thr Ser Ser
85 90 95
Phe Asn Phe Thr Ile Thr Ala Ser Gln Val Val Asp Ser Ala Val Tyr
100 105 110
Phe Cys Ala Leu Ser Glu Ala Ser Gly Arg Asp Asp Lys Ile Ile Phe
115 120 125
Gly Lys Gly Thr Arg Leu His Ile Leu Pro Asn Ile Gln Asn Pro Asp
130 135 140
Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val
145 150 155 160
Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys
165 170 175
Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser
180 185 190
Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp
195 200 205
Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr
210 215 220
Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys
225 230 235 240
Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile
245 250 255
Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met
260 265 270
Thr Leu Arg Leu Trp Ser Ser
275
<210> 22
<211> 309
<212> PRT
<213> Human
<400> 22
Met Leu Leu Leu Leu Leu Leu Leu Gly Pro Gly Ser Gly Leu Gly Ala
1 5 10 15
Val Val Ser Gln His Pro Ser Trp Val Ile Cys Lys Ser Gly Thr Ser
20 25 30
Val Lys Ile Glu Cys Arg Ser Leu Asp Phe Gln Ala Thr Thr Met Phe
35 40 45
Trp Tyr Arg Gln Phe Pro Lys Gln Ser Leu Met Leu Met Ala Thr Ser
50 55 60
Asn Glu Gly Ser Lys Ala Thr Tyr Glu Gln Gly Val Glu Lys Asp Lys
65 70 75 80
Phe Leu Ile Asn His Ala Ser Leu Thr Leu Ser Thr Leu Thr Val Thr
85 90 95
Ser Ala His Pro Glu Asp Ser Ser Phe Tyr Ile Cys Ser Ala Arg Val
100 105 110
Asp Phe Asp Arg Asp Glu Gln Phe Phe Gly Pro Gly Thr Arg Leu Thr
115 120 125
Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe
130 135 140
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
145 150 155 160
Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp
165 170 175
Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro
180 185 190
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
195 200 205
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
210 215 220
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
225 230 235 240
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
245 250 255
Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val
260 265 270
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
275 280 285
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
290 295 300
Lys Asp Ser Arg Gly
305
<210> 23
<211> 822
<212> DNA
<213> Human
<400> 23
atggaaactc tcctgggagt gtctttggtg attctatggc ttcaactggc tagggtgaac 60
agtcaacagg gagaagagga tcctcaggcc ttgagcatcc aggagggtga aaatgccacc 120
atgaactgca gttacaaaac tagtataaac aatttacagt ggtatagaca aaattcaggt 180
agaggccttg tccacctaat tttaatacgt tcaaatgaaa gagagaaaca cagtggaaga 240
ttaagagtca cgcttgacac ttccaagaaa agcagttcct tgttgatcac ggcttcccgg 300
gcagcagaca ctgcttctta cttctgtgct acggacgcag agtataacaa tgccagactc 360
atgtttggag atggaactca gctggtggtg aagcccaata tccagaaccc tgaccctgcc 420
gtgtaccagt tgagagactc taaatccagt gacaagtctg tctgcctatt caccgatttt 480
gattctcaaa caaatgtgtc acaaagtaag gattctgatg tgtatatcac agacaaaact 540
gtgctagaca tgaggtctat ggacttcaag agcaacagtg ctgtggcctg gagcaacaaa 600
tctgactttg catgtgcaaa cgccttcaac aacagcatta ttccagaaga caccttcttc 660
cccagcccag aaagttcctg tgatgtcaag ctggtcgaga aaagctttga aacagatacg 720
aacctaaact ttcaaaacct gtcagtgatt gggttccgaa tcctcctcct gaaagtggcc 780
gggtttaatc tgctcatgac gctgcggctg tggtccagct ga 822
<210> 24
<211> 939
<212> DNA
<213> Human
<400> 24
atgggcacca gcctcctctg ctggatggcc ctgtgtctcc tgggggcaga tcacgcagat 60
actggagtct cccaggaccc cagacacaag atcacaaaga ggggacagaa tgtaactttc 120
aggtgtgatc caatttctga acacaaccgc ctttattggt accgacagac cctggggcag 180
ggcccagagt ttctgactta cttccagaat gaagctcaac tagaaaaatc aaggctgctc 240
agtgatcggt tctctgcaga gaggcctaag ggatctttct ccaccttgga gatccagcgc 300
acagagcagg gggactcggc catgtatctc tgtgccagca gcatggtagc tggggccaac 360
gtcctgactt tcggggccgg cagcaggctg accgtgctgg aggacctgaa aaacgtgttc 420
ccacccgagg tcgctgtgtt tgagccatca gaagcagaga tctcccacac ccaaaaggcc 480
acactggtgt gcctggccac aggcttctac cccgaccacg tggagctgag ctggtgggtg 540
aatgggaagg aggtgcacag tggggtcagc acagacccgc agcccctcaa ggagcagccc 600
gccctcaatg actccagata ctgcctgagc agccgcctga gggtctcggc caccttctgg 660
cagaaccccc gcaaccactt ccgctgtcaa gtccagttct acgggctctc ggagaatgac 720
gagtggaccc aggatagggc caaacctgtc acccagatcg tcagcgccga ggcctggggt 780
agagcagact gtggcttcac ctccgagtct taccagcaag gggtcctgtc tgccaccatc 840
ctctatgaga tcttgctagg gaaggccacc ttgtatgccg tgctggtcag tgccctcgtg 900
ctgatggcca tggtcaagag aaaggattcc agaggctag 939
<210> 25
<211> 273
<212> PRT
<213> Human
<400> 25
Met Glu Thr Leu Leu Gly Val Ser Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ala Arg Val Asn Ser Gln Gln Gly Glu Glu Asp Pro Gln Ala Leu Ser
20 25 30
Ile Gln Glu Gly Glu Asn Ala Thr Met Asn Cys Ser Tyr Lys Thr Ser
35 40 45
Ile Asn Asn Leu Gln Trp Tyr Arg Gln Asn Ser Gly Arg Gly Leu Val
50 55 60
His Leu Ile Leu Ile Arg Ser Asn Glu Arg Glu Lys His Ser Gly Arg
65 70 75 80
Leu Arg Val Thr Leu Asp Thr Ser Lys Lys Ser Ser Ser Leu Leu Ile
85 90 95
Thr Ala Ser Arg Ala Ala Asp Thr Ala Ser Tyr Phe Cys Ala Thr Asp
100 105 110
Ala Glu Tyr Asn Asn Ala Arg Leu Met Phe Gly Asp Gly Thr Gln Leu
115 120 125
Val Val Lys Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
130 135 140
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
145 150 155 160
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
165 170 175
Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
180 185 190
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
195 200 205
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
210 215 220
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
225 230 235 240
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
245 250 255
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
260 265 270
Ser
<210> 26
<211> 312
<212> PRT
<213> Human
<400> 26
Met Gly Thr Ser Leu Leu Cys Trp Met Ala Leu Cys Leu Leu Gly Ala
1 5 10 15
Asp His Ala Asp Thr Gly Val Ser Gln Asp Pro Arg His Lys Ile Thr
20 25 30
Lys Arg Gly Gln Asn Val Thr Phe Arg Cys Asp Pro Ile Ser Glu His
35 40 45
Asn Arg Leu Tyr Trp Tyr Arg Gln Thr Leu Gly Gln Gly Pro Glu Phe
50 55 60
Leu Thr Tyr Phe Gln Asn Glu Ala Gln Leu Glu Lys Ser Arg Leu Leu
65 70 75 80
Ser Asp Arg Phe Ser Ala Glu Arg Pro Lys Gly Ser Phe Ser Thr Leu
85 90 95
Glu Ile Gln Arg Thr Glu Gln Gly Asp Ser Ala Met Tyr Leu Cys Ala
100 105 110
Ser Ser Met Val Ala Gly Ala Asn Val Leu Thr Phe Gly Ala Gly Ser
115 120 125
Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 27
<211> 819
<212> DNA
<213> Human
<400> 27
atgatgaaat ccttgagagt tttactagtg atcctgtggc ttcagttgag ctgggtttgg 60
agccaacaga aggaggtgga gcagaattct ggacccctca gtgttccaga gggagccatt 120
gcctctctca actgcactta cagtgaccga ggttcccagt ccttcttctg gtacagacaa 180
tattctggga aaagccctga gttgataatg ttcatatact ccaatggtga caaagaagat 240
ggaaggttta cagcacagct caataaagcc agccagtatg tttctctgct catcagagac 300
tcccagccca gtgattcagc cacctacctc tgtgccgtgg gtgctacaaa caagctcatc 360
tttggaactg gcactctgct tgctgtccag ccaaatatcc agaaccctga ccctgccgtg 420
taccagctga gagactctaa atccagtgac aagtctgtct gcctattcac cgattttgat 480
tctcaaacaa atgtgtcaca aagtaaggat tctgatgtgt atatcacaga caaaactgtg 540
ctagacatga ggtctatgga cttcaagagc aacagtgctg tggcctggag caacaaatct 600
gactttgcat gtgcaaacgc cttcaacaac agcattattc cagaagacac cttcttcccc 660
agcccagaaa gttcctgtga tgtcaagctg gtcgagaaaa gctttgaaac agatacgaac 720
ctaaactttc aaaacctgtc agtgattggg ttccgaatcc tcctcctgaa agtggccggg 780
tttaatctgc tcatgacgct gcggctgtgg tccagctga 819
<210> 28
<211> 930
<212> DNA
<213> Human
<400> 28
atggtttcca ggcttctcag tttagtgtcc ctttgtctcc tgggagcaaa gcacatagaa 60
gctggagtta ctcagttccc cagccacagc gtaatagaga agggccagac tgtgactctg 120
agatgtgacc caatttctgg acatgataat ctttattggt atcgacgtgt tatgggaaaa 180
gaaataaaat ttctgttaca ttttgtgaaa gagtctaaac aggatgagtc cggtatgccc 240
aacaatcgat tcttagctga aaggactgga gggacgtatt ctactctgaa ggtgcagcct 300
gcagaactgg aggattctgg agtttatttc tgtgccagca gccaagcgta cggcactgaa 360
gctttctttg gacaaggcac cagactcaca gttgtagagg acctgaacaa ggtgttccca 420
cccgaggtcg ctgtgtttga gccatcagaa gcagagatct cccacaccca aaaggccaca 480
ctggtgtgcc tggccacagg cttcttccct gaccacgtgg agctgagctg gtgggtgaat 540
gggaaggagg tgcacagtgg ggtcagcacg gacccgcagc ccctcaagga gcagcccgcc 600
ctcaatgact ccagatactg cctgagcagc cgcctgaggg tctcggccac cttctggcag 660
aacccccgca accacttccg ctgtcaagtc cagttctacg ggctctcgga gaatgacgag 720
tggacccagg atagggccaa acccgtcacc cagatcgtca gcgccgaggc ctggggtaga 780
gcagactgtg gctttacctc ggtgtcctac cagcaagggg tcctgtctgc caccatcctc 840
tatgagatcc tgctagggaa ggccaccctg tatgctgtgc tggtcagcgc ccttgtgttg 900
atggccatgg tcaagagaaa ggatttctga 930
<210> 29
<211> 272
<212> PRT
<213> Human
<400> 29
Met Met Lys Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ser Trp Val Trp Ser Gln Gln Lys Glu Val Glu Gln Asn Ser Gly Pro
20 25 30
Leu Ser Val Pro Glu Gly Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser
35 40 45
Asp Arg Gly Ser Gln Ser Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys
50 55 60
Ser Pro Glu Leu Ile Met Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp
65 70 75 80
Gly Arg Phe Thr Ala Gln Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu
85 90 95
Leu Ile Arg Asp Ser Gln Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala
100 105 110
Val Gly Ala Thr Asn Lys Leu Ile Phe Gly Thr Gly Thr Leu Leu Ala
115 120 125
Val Gln Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg
130 135 140
Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp
145 150 155 160
Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr
165 170 175
Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser
180 185 190
Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe
195 200 205
Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser
210 215 220
Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn
225 230 235 240
Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu
245 250 255
Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265 270
<210> 30
<211> 309
<212> PRT
<213> Human
<400> 30
Met Val Ser Arg Leu Leu Ser Leu Val Ser Leu Cys Leu Leu Gly Ala
1 5 10 15
Lys His Ile Glu Ala Gly Val Thr Gln Phe Pro Ser His Ser Val Ile
20 25 30
Glu Lys Gly Gln Thr Val Thr Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Asp Asn Leu Tyr Trp Tyr Arg Arg Val Met Gly Lys Glu Ile Lys Phe
50 55 60
Leu Leu His Phe Val Lys Glu Ser Lys Gln Asp Glu Ser Gly Met Pro
65 70 75 80
Asn Asn Arg Phe Leu Ala Glu Arg Thr Gly Gly Thr Tyr Ser Thr Leu
85 90 95
Lys Val Gln Pro Ala Glu Leu Glu Asp Ser Gly Val Tyr Phe Cys Ala
100 105 110
Ser Ser Gln Ala Tyr Gly Thr Glu Ala Phe Phe Gly Gln Gly Thr Arg
115 120 125
Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Phe
305
<210> 31
<211> 843
<212> DNA
<213> Human
<400> 31
atggccatgc tcctgggggc atcagtgctg attctgtggc ttcagccaga ctgggtaaac 60
agtcaacaga agaatgatga ccagcaagtt aagcaaaatt caccatccct gagcgtccag 120
gaaggaagaa tttctattct gaactgtgac tatactaaca gcatgtttga ttatttccta 180
tggtacaaaa aataccctgc tgaaggtcct acattcctga tatctataag ttccattaag 240
gataaaaatg aagatggaag attcactgtc ttcttaaaca aaagtgccaa gcacctctct 300
ctgcacattg tgccctccca gcctggagac tctgcagtgt acttctgtgc agcaagcccc 360
tctggcaaca caggcaaact aatctttggg caagggacaa ctttacaagt aaaaccagat 420
atccagaagc ctgaccctgc cgtgtaccag ctgagagact ctaaatccag tgacaagtct 480
gtctgcctat tcaccgattt tgattctcaa acaaatgtgt cacaaagtaa ggattctgat 540
gtgtatatca cagacaaaac tgtgctagac atgaggtcta tggacttcaa gagcaacagt 600
gctgtggcct ggagcaacaa atctgacttt gcatgtgcaa acgccttcaa caacagcatt 660
attccagaag acaccttctt ccccagccca gaaagttcct gtgatgtcaa gctggtcgag 720
aaaagctttg aaacagatac gaacctaaac tttcaaaacc tgtcagtgat tgggttccga 780
atcctcctcc tgaaagtggc cgggtttaat ctgctcatga cgctgcggct gtggtccagc 840
tga 843
<210> 32
<211> 933
<212> DNA
<213> Human
<400> 32
atgggctcca ggctgctctg ttgggtgctg ctttgtctcc tgggagcagg cccagtaaag 60
gctggagtca ctcaaactcc aagatatctg atcaaaacga gaggacagca agtgacactg 120
agctgctccc ctatctctgg gcataggagt gtatcctggt accaacagac cccaggacag 180
ggccttcagt tcctctttga atacttcagt gagacacaga gaaacaaagg aaacttccct 240
ggtcgattct cagggcgcca gttctctaac tctcgctctg agatgaatgt gagcaccttg 300
gagctggggg actcggccct ttatctttgc gccagcagcc cagacagggc catgaacact 360
gaagctttct ttggacaagg caccagactc acagttgtag aggacctgaa caaggtgttc 420
ccacccgagg tcgctgtgtt tgagccatca gaagcagaga tctcccacac ccaaaaggcc 480
acactggtgt gcctggccac aggcttcttc cctgaccacg tggagctgag ctggtgggtg 540
aatgggaagg aggtgcacag tggggtcagc acggacccgc agcccctcaa ggagcagccc 600
gccctcaatg actccagata ctgcctgagc agccgcctga gggtctcggc caccttctgg 660
cagaaccccc gcaaccactt ccgctgtcaa gtccagttct acgggctctc ggagaatgac 720
gagtggaccc aggatagggc caaacccgtc acccagatcg tcagcgccga ggcctggggt 780
agagcagact gtggctttac ctcggtgtcc taccagcaag gggtcctgtc tgccaccatc 840
ctctatgaga tcctgctagg gaaggccacc ctgtatgctg tgctggtcag cgcccttgtg 900
ttgatggcca tggtcaagag aaaggatttc tga 933
<210> 33
<211> 280
<212> PRT
<213> Human
<400> 33
Met Ala Met Leu Leu Gly Ala Ser Val Leu Ile Leu Trp Leu Gln Pro
1 5 10 15
Asp Trp Val Asn Ser Gln Gln Lys Asn Asp Asp Gln Gln Val Lys Gln
20 25 30
Asn Ser Pro Ser Leu Ser Val Gln Glu Gly Arg Ile Ser Ile Leu Asn
35 40 45
Cys Asp Tyr Thr Asn Ser Met Phe Asp Tyr Phe Leu Trp Tyr Lys Lys
50 55 60
Tyr Pro Ala Glu Gly Pro Thr Phe Leu Ile Ser Ile Ser Ser Ile Lys
65 70 75 80
Asp Lys Asn Glu Asp Gly Arg Phe Thr Val Phe Leu Asn Lys Ser Ala
85 90 95
Lys His Leu Ser Leu His Ile Val Pro Ser Gln Pro Gly Asp Ser Ala
100 105 110
Val Tyr Phe Cys Ala Ala Ser Pro Ser Gly Asn Thr Gly Lys Leu Ile
115 120 125
Phe Gly Gln Gly Thr Thr Leu Gln Val Lys Pro Asp Ile Gln Lys Pro
130 135 140
Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser
145 150 155 160
Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser
165 170 175
Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg
180 185 190
Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser
195 200 205
Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp
210 215 220
Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu
225 230 235 240
Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val
245 250 255
Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu
260 265 270
Met Thr Leu Arg Leu Trp Ser Ser
275 280
<210> 34
<211> 310
<212> PRT
<213> Human
<400> 34
Met Gly Ser Arg Leu Leu Cys Trp Val Leu Leu Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Lys Ala Gly Val Thr Gln Thr Pro Arg Tyr Leu Ile Lys
20 25 30
Thr Arg Gly Gln Gln Val Thr Leu Ser Cys Ser Pro Ile Ser Gly His
35 40 45
Arg Ser Val Ser Trp Tyr Gln Gln Thr Pro Gly Gln Gly Leu Gln Phe
50 55 60
Leu Phe Glu Tyr Phe Ser Glu Thr Gln Arg Asn Lys Gly Asn Phe Pro
65 70 75 80
Gly Arg Phe Ser Gly Arg Gln Phe Ser Asn Ser Arg Ser Glu Met Asn
85 90 95
Val Ser Thr Leu Glu Leu Gly Asp Ser Ala Leu Tyr Leu Cys Ala Ser
100 105 110
Ser Pro Asp Arg Ala Met Asn Thr Glu Ala Phe Phe Gly Gln Gly Thr
115 120 125
Arg Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe
305 310
<210> 35
<211> 825
<212> DNA
<213> Human
<400> 35
atggcctctg cacccatctc gatgcttgcg atgctcttca cattgagtgg gctgagagct 60
cagtcagtgg ctcagccgga agatcaggtc aacgttgctg aagggaatcc tctgactgtg 120
aaatgcacct attcagtctc tggaaaccct tatctttttt ggtatgttca ataccccaac 180
cgaggcctcc agttccttct gaaatacatc acaggggata acctggttaa aggcagctat 240
ggctttgaag ctgaatttaa caagagccaa acctccttcc acctgaagaa accatctgcc 300
cttgtgagcg actccgcttt gtacttctgt gctgtgagag acagggagag agatgacaag 360
atcatctttg gaaaagggac acgacttcat attctcccca atatccagaa gcctgaccct 420
gccgtgtacc agctgagaga ctctaaatcc agtgacaagt ctgtctgcct attcaccgat 480
tttgattctc aaacaaatgt gtcacaaagt aaggattctg atgtgtatat cacagacaaa 540
actgtgctag acatgaggtc tatggacttc aagagcaaca gtgctgtggc ctggagcaac 600
aaatctgact ttgcatgtgc aaacgccttc aacaacagca ttattccaga agacaccttc 660
ttccccagcc cagaaagttc ctgtgatgtc aagctggtcg agaaaagctt tgaaacagat 720
acgaacctaa actttcaaaa cctgtcagtg attgggttcc gaatcctcct cctgaaagtg 780
gccgggttta atctgctcat gacgctgcgg ctgtggtcca gctga 825
<210> 36
<211> 936
<212> DNA
<213> Human
<400> 36
atggatacct ggctcgtatg ctgggcaatt tttagtctct tgaaagcagg actcacagaa 60
cctgaagtca cccagactcc cagccatcag gtcacacaga tgggacagga agtgatcttg 120
cgctgtgtcc ccatctctaa tcacttatac ttctattggt acagacaaat cttggggcag 180
aaagtcgagt ttctggtttc cttttataat aatgaaatct cagagaagtc tgaaatattc 240
gatgatcaat tctcagttga aaggcctgat ggatcaaatt tcactctgaa gatccggtcc 300
acaaagctgg aggactcagc catgtacttc tgtgccagca gagcggagat cacagatacg 360
cagtattttg gcccaggcac ccggctgaca gtgctcgagg acctgaaaaa cgtgttccca 420
cccgaggtcg ctgtgtttga gccatcagaa gcagagatct cccacaccca aaaggccaca 480
ctggtgtgcc tggccacagg cttctacccc gaccacgtgg agctgagctg gtgggtgaat 540
gggaaggagg tgcacagtgg ggtcagcaca gacccgcagc ccctcaagga gcagcccgcc 600
ctcaatgact ccagatactg cctgagcagc cgcctgaggg tctcggccac cttctggcag 660
aacccccgca accacttccg ctgtcaagtc cagttctacg ggctctcgga gaatgacgag 720
tggacccagg atagggccaa acctgtcacc cagatcgtca gcgccgaggc ctggggtaga 780
gcagactgtg gcttcacctc cgagtcttac cagcaagggg tcctgtctgc caccatcctc 840
tatgagatct tgctagggaa ggccaccttg tatgccgtgc tggtcagtgc cctcgtgctg 900
atggccatgg tcaagagaaa ggattccaga ggctag 936
<210> 37
<211> 274
<212> PRT
<213> Human
<400> 37
Met Ala Ser Ala Pro Ile Ser Met Leu Ala Met Leu Phe Thr Leu Ser
1 5 10 15
Gly Leu Arg Ala Gln Ser Val Ala Gln Pro Glu Asp Gln Val Asn Val
20 25 30
Ala Glu Gly Asn Pro Leu Thr Val Lys Cys Thr Tyr Ser Val Ser Gly
35 40 45
Asn Pro Tyr Leu Phe Trp Tyr Val Gln Tyr Pro Asn Arg Gly Leu Gln
50 55 60
Phe Leu Leu Lys Tyr Ile Thr Gly Asp Asn Leu Val Lys Gly Ser Tyr
65 70 75 80
Gly Phe Glu Ala Glu Phe Asn Lys Ser Gln Thr Ser Phe His Leu Lys
85 90 95
Lys Pro Ser Ala Leu Val Ser Asp Ser Ala Leu Tyr Phe Cys Ala Val
100 105 110
Arg Asp Arg Glu Arg Asp Asp Lys Ile Ile Phe Gly Lys Gly Thr Arg
115 120 125
Leu His Ile Leu Pro Asn Ile Gln Lys Pro Asp Pro Ala Val Tyr Gln
130 135 140
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
145 150 155 160
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
165 170 175
Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
180 185 190
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
195 200 205
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
210 215 220
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
225 230 235 240
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
245 250 255
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
260 265 270
Ser Ser
<210> 38
<211> 311
<212> PRT
<213> Human
<400> 38
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Arg Ala Glu Ile Thr Asp Thr Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 39
<211> 843
<212> DNA
<213> Human
<400> 39
atggccatgc tcctgggggc atcagtgctg attctgtggc ttcagccaga ctgggtaaac 60
agtcaacaga agaatgatga ccagcaagtt aagcaaaatt caccatccct gagcgtccag 120
gaaggaagaa tttctattct gaactgtgac tatactaaca gcatgtttga ttatttccta 180
tggtacaaaa aataccctgc tgaaggtcct acattcctga tatctataag ttccattaag 240
gataaaaatg aagatggaag attcactgtc ttcttaaaca aaagtgccaa gcacctctct 300
ctgcacattg tgccctccca gcctggagac tctgcagtgt acttctgtgc agcaagcgcg 360
ataggaggtg ctgacggact cacctttggc aaagggactc atctaatcat ccagccctat 420
atccagaacc ctgaccctgc cgtgtaccag ctgagagact ctaaatccag tgacaagtct 480
gtctgcctat tcaccgattt tgattctcaa acaaatgtgt cacaaagtaa ggattctgat 540
gtgtatatca cagacaaaac tgtgctagac atgaggtcta tggacttcaa gagcaacagt 600
gctgtggcct ggagcaacaa atctgacttt gcatgtgcaa acgccttcaa caacagcatt 660
attccagaag acaccttctt ccccagccca gaaagttcct gtgatgtcaa gctggtcgag 720
aaaagctttg aaacagatac gaacctaaac tttcaaaacc tgtcagtgat tgggttccga 780
atcctcctcc tgaaagtggc cgggtttaat ctgctcatga cgctgcggct gtggtccagc 840
tga 843
<210> 40
<211> 939
<212> DNA
<213> Human
<400> 40
atgagcctcg ggctcctgtg ctgtggggcc ttttctctcc tgtgggcagg tccagtgaat 60
gctggtgtca ctcagacccc aaaattccgg gtcctgaaga caggacagag catgacactg 120
ctgtgtgccc aggatatgaa ccatgaatac atgtactggt atcgacaaga cccaggcatg 180
gggctgaggc tgattcatta ctcagttggt gagggtacaa ctgccaaagg agaggtccct 240
gatggctaca atgtctccag attaaaaaaa cagaatttcc tgctggggtt ggagtcggct 300
gctccctccc aaacatctgt gtacttctgt gccagcagta ttcggggaaa aaggtacaat 360
gagcagttct tcgggccagg gacacggctc accgtgctag aggacctgaa aaacgtgttc 420
ccacccgagg tcgctgtgtt tgagccatca gaagcagaga tctcccacac ccaaaaggcc 480
acactggtgt gcctggccac aggcttctac cccgaccacg tggagctgag ctggtgggtg 540
aatgggaagg aggtgcacag tggggtcagc acagacccgc agcccctcaa ggagcagccc 600
gccctcaatg actccagata ctgcctgagc agccgcctga gggtctcggc caccttctgg 660
cagaaccccc gcaaccactt ccgctgtcaa gtccagttct acgggctctc ggagaatgac 720
gagtggaccc aggatagggc caaacctgtc acccagatcg tcagcgccga ggcctggggt 780
agagcagact gtggcttcac ctccgagtct taccagcaag gggtcctgtc tgccaccatc 840
ctctatgaga tcttgctagg gaaggccacc ttgtatgccg tgctggtcag tgccctcgtg 900
ctgatggcca tggtcaagag aaaggattcc agaggctag 939
<210> 41
<211> 280
<212> PRT
<213> Human
<400> 41
Met Ala Met Leu Leu Gly Ala Ser Val Leu Ile Leu Trp Leu Gln Pro
1 5 10 15
Asp Trp Val Asn Ser Gln Gln Lys Asn Asp Asp Gln Gln Val Lys Gln
20 25 30
Asn Ser Pro Ser Leu Ser Val Gln Glu Gly Arg Ile Ser Ile Leu Asn
35 40 45
Cys Asp Tyr Thr Asn Ser Met Phe Asp Tyr Phe Leu Trp Tyr Lys Lys
50 55 60
Tyr Pro Ala Glu Gly Pro Thr Phe Leu Ile Ser Ile Ser Ser Ile Lys
65 70 75 80
Asp Lys Asn Glu Asp Gly Arg Phe Thr Val Phe Leu Asn Lys Ser Ala
85 90 95
Lys His Leu Ser Leu His Ile Val Pro Ser Gln Pro Gly Asp Ser Ala
100 105 110
Val Tyr Phe Cys Ala Ala Ser Ala Ile Gly Gly Ala Asp Gly Leu Thr
115 120 125
Phe Gly Lys Gly Thr His Leu Ile Ile Gln Pro Tyr Ile Gln Asn Pro
130 135 140
Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser
145 150 155 160
Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser
165 170 175
Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg
180 185 190
Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser
195 200 205
Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp
210 215 220
Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu
225 230 235 240
Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val
245 250 255
Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu
260 265 270
Met Thr Leu Arg Leu Trp Ser Ser
275 280
<210> 42
<211> 312
<212> PRT
<213> Human
<400> 42
Met Ser Leu Gly Leu Leu Cys Cys Gly Ala Phe Ser Leu Leu Trp Ala
1 5 10 15
Gly Pro Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Arg Val Leu
20 25 30
Lys Thr Gly Gln Ser Met Thr Leu Leu Cys Ala Gln Asp Met Asn His
35 40 45
Glu Tyr Met Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu
50 55 60
Ile His Tyr Ser Val Gly Glu Gly Thr Thr Ala Lys Gly Glu Val Pro
65 70 75 80
Asp Gly Tyr Asn Val Ser Arg Leu Lys Lys Gln Asn Phe Leu Leu Gly
85 90 95
Leu Glu Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser
100 105 110
Ser Ile Arg Gly Lys Arg Tyr Asn Glu Gln Phe Phe Gly Pro Gly Thr
115 120 125
Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 43
<211> 822
<212> DNA
<213> Human
<400> 43
atggaaactc tcctgggagt gtctttggtg attctatggc ttcaactggc tagggtgaac 60
agtcaacagg gagaagagga tcctcaggcc ttgagcatcc aggagggtga aaatgccacc 120
atgaactgca gttacaaaac tagtataaac aatttacagt ggtatagaca aaattcaggt 180
agaggccttg tccacctaat tttaatacgt tcaaatgaaa gagagaaaca cagtggaaga 240
ttaagagtca cgcttgacac ttccaagaaa agcagttcct tgttgatcac ggcttcccgg 300
gcagcagaca ctgcttctta cttctgtgct acggacgccg tcgtgggtgc tgacggactc 360
acctttggca aagggactca tctaatcatc cagccctata tccagaaccc tgaccctgcc 420
gtgtaccagc tgagagactc taaatccagt gacaagtctg tctgcctatt caccgatttt 480
gattctcaaa caaatgtgtc acaaagtaag gattctgatg tgtatatcac agacaaaact 540
gtgctagaca tgaggtctat ggacttcaag agcaacagtg ctgtggcctg gagcaacaaa 600
tctgactttg catgtgcaaa cgccttcaac aacagcatta ttccagaaga caccttcttc 660
cccagcccag aaagttcctg tgatgtcaag ctggtcgaga aaagctttga aacagatacg 720
aacctaaact ttcaaaacct gtcagtgatt gggttccgaa tcctcctcct gaaagtggcc 780
gggtttaatc tgctcatgac gctgcggctg tggtccagct ga 822
<210> 44
<211> 936
<212> DNA
<213> Human
<400> 44
atgctgctgc ttctgctgct tctggggcca ggctccgggc ttggtgctgt cgtctctcaa 60
catccgagct gggttatctg taagagtgga acctctgtga agatcgagtg ccgttccctg 120
gactttcagg ccacaactat gttttggtat cgtcagttcc cgaaacagag tctcatgctg 180
atggcaactt ccaatgaggg ctccaaggcc acatacgagc aaggcgtcga gaaggacaag 240
tttctcatca accatgcaag cctgaccttg tccactctga cagtgaccag tgcccatcct 300
gaagacagca gcttctacat ctgcagtgct agagaccggg acataggacc cttagatacg 360
cagtattttg gcccaggcac ccggctgaca gtgctcgagg acctgaaaaa cgtgttccca 420
cccgaggtcg ctgtgtttga gccatcagaa gcagagatct cccacaccca aaaggccaca 480
ctggtgtgcc tggccacagg cttctacccc gaccacgtgg agctgagctg gtgggtgaat 540
gggaaggagg tgcacagtgg ggtcagcaca gacccgcagc ccctcaagga gcagcccgcc 600
ctcaatgact ccagatactg cctgagcagc cgcctgaggg tctcggccac cttctggcag 660
aacccccgca accacttccg ctgtcaagtc cagttctacg ggctctcgga gaatgacgag 720
tggacccagg atagggccaa acctgtcacc cagatcgtca gcgccgaggc ctggggtaga 780
gcagactgtg gcttcacctc cgagtcttac cagcaagggg tcctgtctgc caccatcctc 840
tatgagatct tgctagggaa ggccaccttg tatgccgtgc tggtcagtgc cctcgtgctg 900
atggccatgg tcaagagaaa ggattccaga ggctag 936
<210> 45
<211> 273
<212> PRT
<213> Human
<400> 45
Met Glu Thr Leu Leu Gly Val Ser Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ala Arg Val Asn Ser Gln Gln Gly Glu Glu Asp Pro Gln Ala Leu Ser
20 25 30
Ile Gln Glu Gly Glu Asn Ala Thr Met Asn Cys Ser Tyr Lys Thr Ser
35 40 45
Ile Asn Asn Leu Gln Trp Tyr Arg Gln Asn Ser Gly Arg Gly Leu Val
50 55 60
His Leu Ile Leu Ile Arg Ser Asn Glu Arg Glu Lys His Ser Gly Arg
65 70 75 80
Leu Arg Val Thr Leu Asp Thr Ser Lys Lys Ser Ser Ser Leu Leu Ile
85 90 95
Thr Ala Ser Arg Ala Ala Asp Thr Ala Ser Tyr Phe Cys Ala Thr Asp
100 105 110
Ala Val Val Gly Ala Asp Gly Leu Thr Phe Gly Lys Gly Thr His Leu
115 120 125
Ile Ile Gln Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
130 135 140
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
145 150 155 160
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
165 170 175
Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
180 185 190
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
195 200 205
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
210 215 220
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
225 230 235 240
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
245 250 255
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
260 265 270
Ser
<210> 46
<211> 311
<212> PRT
<213> Human
<400> 46
Met Leu Leu Leu Leu Leu Leu Leu Gly Pro Gly Ser Gly Leu Gly Ala
1 5 10 15
Val Val Ser Gln His Pro Ser Trp Val Ile Cys Lys Ser Gly Thr Ser
20 25 30
Val Lys Ile Glu Cys Arg Ser Leu Asp Phe Gln Ala Thr Thr Met Phe
35 40 45
Trp Tyr Arg Gln Phe Pro Lys Gln Ser Leu Met Leu Met Ala Thr Ser
50 55 60
Asn Glu Gly Ser Lys Ala Thr Tyr Glu Gln Gly Val Glu Lys Asp Lys
65 70 75 80
Phe Leu Ile Asn His Ala Ser Leu Thr Leu Ser Thr Leu Thr Val Thr
85 90 95
Ser Ala His Pro Glu Asp Ser Ser Phe Tyr Ile Cys Ser Ala Arg Asp
100 105 110
Arg Asp Ile Gly Pro Leu Asp Thr Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 47
<211> 798
<212> DNA
<213> Human
<400> 47
atgaagttgg tgacaagcat tactgtactc ctatctttgg gtattatggg tgatgctaag 60
accacacagc caaattcaat ggagagtaac gaagaagagc ctgttcactt gccttgtaac 120
cactccacaa tcagtggaac tgattacata cattggtatc gacagcttcc ctcccagggt 180
ccagagtacg tgattcatgg tcttacaagc aatgtgaaca acagaatggc ctctctggca 240
atcgctgaag acagaaagtc cagtaccttg atcctgcacc gtgctacctt gagagatgct 300
gctgtgtact actgcatccc taataacaat gacatgcgct ttggagcagg gaccagactg 360
acagtaaaac caaatatcca gaaccctgac cctgccgtgt accagctgag agactctaaa 420
tccagtgaca agtctgtctg cctattcacc gattttgatt ctcaaacaaa tgtgtcacaa 480
agtaaggatt ctgatgtgta tatcacagac aaaactgtgc tagacatgag gtctatggac 540
ttcaagagca acagtgctgt ggcctggagc aacaaatctg actttgcatg tgcaaacgcc 600
ttcaacaaca gcattattcc agaagacacc ttcttcccca gcccagaaag ttcctgtgat 660
gtcaagctgg tcgagaaaag ctttgaaaca gatacgaacc taaactttca aaacctgtca 720
gtgattgggt tccgaatcct cctcctgaaa gtggccgggt ttaatctgct catgacgctg 780
cggctgtggt ccagctga 798
<210> 48
<211> 933
<212> DNA
<213> Human
<400> 48
atgggctcca ggctgctctg ttgggtgctg ctttgtctcc tgggagcagg cccagtaaag 60
gctggagtca ctcaaactcc aagatatctg atcaaaacga gaggacagca agtgacactg 120
agctgctccc ctatctctgg gcataggagt gtatcctggt accaacagac cccaggacag 180
ggccttcagt tcctctttga atacttcagt gagacacaga gaaacaaagg aaacttccct 240
ggtcgattct cagggcgcca gttctctaac tctcgctctg agatgaatgt gagcaccttg 300
gagctggggg actcggccct ttatctttgc gccagcagct tccccaggga acctaactat 360
ggctacacct tcggttcggg gaccaggtta accgttgtag aggacctgaa caaggtgttc 420
ccacccgagg tcgctgtgtt tgagccatca gaagcagaga tctcccacac ccaaaaggcc 480
acactggtgt gcctggccac aggcttcttc cctgaccacg tggagctgag ctggtgggtg 540
aatgggaagg aggtgcacag tggggtcagc acggacccgc agcccctcaa ggagcagccc 600
gccctcaatg actccagata ctgcctgagc agccgcctga gggtctcggc caccttctgg 660
cagaaccccc gcaaccactt ccgctgtcaa gtccagttct acgggctctc ggagaatgac 720
gagtggaccc aggatagggc caaacccgtc acccagatcg tcagcgccga ggcctggggt 780
agagcagact gtggctttac ctcggtgtcc taccagcaag gggtcctgtc tgccaccatc 840
ctctatgaga tcctgctagg gaaggccacc ctgtatgctg tgctggtcag cgcccttgtg 900
ttgatggcca tggtcaagag aaaggatttc tga 933
<210> 49
<211> 265
<212> PRT
<213> Human
<400> 49
Met Lys Leu Val Thr Ser Ile Thr Val Leu Leu Ser Leu Gly Ile Met
1 5 10 15
Gly Asp Ala Lys Thr Thr Gln Pro Asn Ser Met Glu Ser Asn Glu Glu
20 25 30
Glu Pro Val His Leu Pro Cys Asn His Ser Thr Ile Ser Gly Thr Asp
35 40 45
Tyr Ile His Trp Tyr Arg Gln Leu Pro Ser Gln Gly Pro Glu Tyr Val
50 55 60
Ile His Gly Leu Thr Ser Asn Val Asn Asn Arg Met Ala Ser Leu Ala
65 70 75 80
Ile Ala Glu Asp Arg Lys Ser Ser Thr Leu Ile Leu His Arg Ala Thr
85 90 95
Leu Arg Asp Ala Ala Val Tyr Tyr Cys Ile Pro Asn Asn Asn Asp Met
100 105 110
Arg Phe Gly Ala Gly Thr Arg Leu Thr Val Lys Pro Asn Ile Gln Asn
115 120 125
Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys
130 135 140
Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln
145 150 155 160
Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met
165 170 175
Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys
180 185 190
Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu
195 200 205
Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val
210 215 220
Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser
225 230 235 240
Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu
245 250 255
Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265
<210> 50
<211> 310
<212> PRT
<213> Human
<400> 50
Met Gly Ser Arg Leu Leu Cys Trp Val Leu Leu Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Lys Ala Gly Val Thr Gln Thr Pro Arg Tyr Leu Ile Lys
20 25 30
Thr Arg Gly Gln Gln Val Thr Leu Ser Cys Ser Pro Ile Ser Gly His
35 40 45
Arg Ser Val Ser Trp Tyr Gln Gln Thr Pro Gly Gln Gly Leu Gln Phe
50 55 60
Leu Phe Glu Tyr Phe Ser Glu Thr Gln Arg Asn Lys Gly Asn Phe Pro
65 70 75 80
Gly Arg Phe Ser Gly Arg Gln Phe Ser Asn Ser Arg Ser Glu Met Asn
85 90 95
Val Ser Thr Leu Glu Leu Gly Asp Ser Ala Leu Tyr Leu Cys Ala Ser
100 105 110
Ser Phe Pro Arg Glu Pro Asn Tyr Gly Tyr Thr Phe Gly Ser Gly Thr
115 120 125
Arg Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe
305 310
<210> 51
<211> 780
<212> DNA
<213> Human
<400> 51
atggcccaga cagtcactca gtctcaacca gagatgtctg tgcaggaggc agagactgtg 60
accctgagtt gcacatatga caccagtgag aataattatt atttgttctg gtacaagcag 120
cctcccagca ggcagatgat tctcgttatt cgccaagaag cttataagca acagaatgca 180
acggagaatc gtttctctgt gaacttccag aaagcagcca aatccttcag tctcaagatc 240
tcagactcac agctggggga cactgcgatg tatttctgtg ctttctcgag agggagtgga 300
ggtagcaact ataaactgac atttggaaaa ggaactctct taaccgtgaa tccaaatatc 360
cagaaccctg accctgccgt gtaccagctg agagactcta aatccagtga caagtctgtc 420
tgcctattca ccgattttga ttctcaaaca aatgtgtcac aaagtaagga ttctgatgtg 480
tatatcacag acaaaactgt gctagacatg aggtctatgg acttcaagag caacagtgct 540
gtggcctgga gcaacaaatc tgactttgca tgtgcaaacg ccttcaacaa cagcattatt 600
ccagaagaca ccttcttccc cagcccagaa agttcctgtg atgtcaagct ggtcgagaaa 660
agctttgaaa cagatacgaa cctaaacttt caaaacctgt cagtgattgg gttccgaatc 720
ctcctcctga aagtggccgg gtttaatctg ctcatgacgc tgcggctgtg gtccagctga 780
<210> 52
<211> 942
<212> DNA
<213> Human
<400> 52
atgggcacca ggctcctctt ctgggtggcc ttctgtctcc tgggggcaga tcacacagga 60
gctggagtct cccagtcccc cagtaacaag gtcacagaga agggaaagga tgtagagctc 120
aggtgtgatc caatttcagg tcatactgcc ctttactggt accgacagag cctggggcag 180
ggcctggagt ttttaattta cttccaaggc aacagtgcac cagacaaatc agggctgccc 240
agtgatcgct tctctgcaga gaggactggg ggatccgtct ccactctgac gatccagcgc 300
acacagcagg aggactcggc cgtgtatctc tgtgccagca gcttagtccc cgacagtgcc 360
tacgagcagt acttcgggcc gggcaccagg ctcacggtca cagaggacct gaaaaacgtg 420
ttcccacccg aggtcgctgt gtttgagcca tcagaagcag agatctccca cacccaaaag 480
gccacactgg tatgcctggc cacaggcttc taccccgacc acgtggagct gagctggtgg 540
gtgaatggga aggaggtgca cagtggggtc agcacggacc cgcagcccct caaggagcag 600
cccgccctca atgactccag atactgcctg agcagccgcc tgagggtctc ggccaccttc 660
tggcagaacc cccgcaacca cttccgctgt caagtccagt tctacgggct ctcggagaat 720
gacgagtgga cccaggatag ggccaaacct gtcacccaga tcgtcagcgc cgaggcctgg 780
ggtagagcag actgtggctt cacctccgag tcttaccagc aaggggtcct gtctgccacc 840
atcctctatg agatcttgct agggaaggcc accttgtatg ccgtgctggt cagtgccctc 900
gtgctgatgg ccatggtcaa gagaaaggat tccagaggct ag 942
<210> 53
<211> 259
<212> PRT
<213> Human
<400> 53
Met Ala Gln Thr Val Thr Gln Ser Gln Pro Glu Met Ser Val Gln Glu
1 5 10 15
Ala Glu Thr Val Thr Leu Ser Cys Thr Tyr Asp Thr Ser Glu Asn Asn
20 25 30
Tyr Tyr Leu Phe Trp Tyr Lys Gln Pro Pro Ser Arg Gln Met Ile Leu
35 40 45
Val Ile Arg Gln Glu Ala Tyr Lys Gln Gln Asn Ala Thr Glu Asn Arg
50 55 60
Phe Ser Val Asn Phe Gln Lys Ala Ala Lys Ser Phe Ser Leu Lys Ile
65 70 75 80
Ser Asp Ser Gln Leu Gly Asp Thr Ala Met Tyr Phe Cys Ala Phe Ser
85 90 95
Arg Gly Ser Gly Gly Ser Asn Tyr Lys Leu Thr Phe Gly Lys Gly Thr
100 105 110
Leu Leu Thr Val Asn Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
115 120 125
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
130 135 140
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
145 150 155 160
Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys
165 170 175
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
180 185 190
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
195 200 205
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
210 215 220
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
225 230 235 240
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
245 250 255
Trp Ser Ser
<210> 54
<211> 313
<212> PRT
<213> Human
<400> 54
Met Gly Thr Arg Leu Leu Phe Trp Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Asp His Thr Gly Ala Gly Val Ser Gln Ser Pro Ser Asn Lys Val Thr
20 25 30
Glu Lys Gly Lys Asp Val Glu Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Thr Ala Leu Tyr Trp Tyr Arg Gln Ser Leu Gly Gln Gly Leu Glu Phe
50 55 60
Leu Ile Tyr Phe Gln Gly Asn Ser Ala Pro Asp Lys Ser Gly Leu Pro
65 70 75 80
Ser Asp Arg Phe Ser Ala Glu Arg Thr Gly Gly Ser Val Ser Thr Leu
85 90 95
Thr Ile Gln Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala
100 105 110
Ser Ser Leu Val Pro Asp Ser Ala Tyr Glu Gln Tyr Phe Gly Pro Gly
115 120 125
Thr Arg Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 55
<211> 828
<212> DNA
<213> Human
<400> 55
atgctcctgc tgctcgtccc agtgctcgag gtgattttta ccctgggagg aaccagagcc 60
cagtcggtga cccagcttgg cagccacgtc tctgtctctg agggagccct ggttctgctg 120
aggtgcaact actcatcgtc tgttccacca tatctcttct ggtatgtgca ataccccaac 180
caaggactcc agcttctcct gaagcacaca acaggggcca ccctggttaa aggcatcaac 240
ggttttgagg ctgaatttaa gaagagtgaa acctccttcc acctgacgaa accctcagcc 300
catatgagcg acgcggctga gtacttctgt gctgtgagtg attctagggc tgcaggcaac 360
aagctaactt ttggaggagg aaccagggtg ctagttaaac caaatatcca gaaccctgac 420
cctgccgtgt accagctgag agactctaaa tccagtgaca agtctgtctg cctattcacc 480
gattttgatt ctcaaacaaa tgtgtcacaa agtaaggatt ctgatgtgta tatcacagac 540
aaaactgtgc tagacatgag gtctatggac ttcaagagca acagtgctgt ggcctggagc 600
aacaaatctg actttgcatg tgcaaacgcc ttcaacaaca gcattattcc agaagacacc 660
ttcttcccca gcccagaaag ttcctgtgat gtcaagctgg tcgagaaaag ctttgaaaca 720
gatacgaacc taaactttca aaacctgtca gtgattgggt tccgaatcct cctcctgaaa 780
gtggccgggt ttaatctgct catgacgctg cggctgtggt ccagctga 828
<210> 56
<211> 936
<212> DNA
<213> Human
<400> 56
atgggaatca ggctcctctg tcgtgtggcc ttttgtttcc tggctgtagg cctcgtagat 60
gtgaaagtaa cccagagctc gagatatcta gtcaaaagga cgggagagaa agtttttctg 120
gaatgtgtcc aggatatgga ccatgaaaat atgttctggt atcgacaaga cccaggtctg 180
gggctacggc tgatctattt ctcatatgat gttaaaatga aagaaaaagg agatattcct 240
gaggggtaca gtgtctctag agagaagaag gagcgcttct ccctgattct ggagtccgcc 300
agcaccaacc agacatctat gtacctctgt gccagcagat tccccgggac agcctataat 360
tcacccctcc actttgggaa tgggaccagg ctcactgtga cagaggacct gaacaaggtg 420
ttcccacccg aggtcgctgt gtttgagcca tcagaagcag agatctccca cacccaaaag 480
gccacactgg tgtgcctggc cacaggcttc ttccctgacc acgtggagct gagctggtgg 540
gtgaatggga aggaggtgca cagtggggtc agcacggacc cgcagcccct caaggagcag 600
cccgccctca atgactccag atactgcctg agcagccgcc tgagggtctc ggccaccttc 660
tggcagaacc cccgcaacca cttccgctgt caagtccagt tctacgggct ctcggagaat 720
gacgagtgga cccaggatag ggccaaaccc gtcacccaga tcgtcagcgc cgaggcctgg 780
ggtagagcag actgtggctt tacctcggtg tcctaccagc aaggggtcct gtctgccacc 840
atcctctatg agatcctgct agggaaggcc accctgtatg ctgtgctggt cagcgccctt 900
gtgttgatgg ccatggtcaa gagaaaggat ttctga 936
<210> 57
<211> 275
<212> PRT
<213> Human
<400> 57
Met Leu Leu Leu Leu Val Pro Val Leu Glu Val Ile Phe Thr Leu Gly
1 5 10 15
Gly Thr Arg Ala Gln Ser Val Thr Gln Leu Gly Ser His Val Ser Val
20 25 30
Ser Glu Gly Ala Leu Val Leu Leu Arg Cys Asn Tyr Ser Ser Ser Val
35 40 45
Pro Pro Tyr Leu Phe Trp Tyr Val Gln Tyr Pro Asn Gln Gly Leu Gln
50 55 60
Leu Leu Leu Lys His Thr Thr Gly Ala Thr Leu Val Lys Gly Ile Asn
65 70 75 80
Gly Phe Glu Ala Glu Phe Lys Lys Ser Glu Thr Ser Phe His Leu Thr
85 90 95
Lys Pro Ser Ala His Met Ser Asp Ala Ala Glu Tyr Phe Cys Ala Val
100 105 110
Ser Asp Ser Arg Ala Ala Gly Asn Lys Leu Thr Phe Gly Gly Gly Thr
115 120 125
Arg Val Leu Val Lys Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
130 135 140
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
145 150 155 160
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
165 170 175
Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys
180 185 190
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
195 200 205
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
210 215 220
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
225 230 235 240
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
245 250 255
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
260 265 270
Trp Ser Ser
275
<210> 58
<211> 311
<212> PRT
<213> Human
<400> 58
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Arg Phe Pro Gly Thr Ala Tyr Asn Ser Pro Leu His Phe Gly Asn Gly
115 120 125
Thr Arg Leu Thr Val Thr Glu Asp Leu Asn Lys Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Phe
305 310
<210> 59
<211> 4
<212> PRT
<213> Artificial Sequence
<220>
<223> Furin protease recognition site
<400> 59
Arg Ala Lys Arg
1
<210> 60
<211> 4
<212> PRT
<213> Artificial Sequence
<220>
<223> linker sequence
<400> 60
Ser Gly Ser Gly
1
<210> 61
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> DP4 peptide sequence
<400> 61
Ser Leu Leu Met Trp Ile Thr Gln Cys Phe Leu Pro Val Phe
1 5 10
<210> 62
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> modified DP4
<400> 62
Leu Leu Met Trp Ile Thr
1 5
<210> 63
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> NY-ESO-1 sequence 157-170
<400> 63
Ser Leu Leu Met Trp Ile Thr Gln Cys Phe Leu Pro Val Phe
1 5 10
<210> 64
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> NY-ESO-1 sequence sequence 95-106
<400> 64
Pro Phe Ala Thr Pro Met Glu Ala Glu Leu Ala Arg
1 5 10
<210> 65
<211> 834
<212> DNA
<213> Human
<400> 65
atgatgaaat ccttgagagt tttactagtg atcctgtggc ttcagttgag ctgggtttgg 60
agccaacaga aggaggtgga gcagaattct ggacccctca gtgttccaga gggagccatt 120
gcctctctca actgcactta cagtgaccga ggttcccagt ccttcttctg gtacagacaa 180
tattctggga aaagccctga gttgataatg tccatatact ccaatggtga caaagaagat 240
ggaaggttta cagcacagct caataaagcc agccagtatg tttctctgct catcagagac 300
tcccagccca gtgattcagc cacctacctc tgtgccgtgg ggggacttac ctctagcaac 360
acaggcaaac taatctttgg gcaagggaca actttacaag taaaaccaga tatccagaac 420
cctgaccctg ccgtgtacca gctgagagac tctaaatcca gtgacaagtc tgtctgccta 480
ttcaccgatt ttgattctca aacaaatgtg tcacaaagta aggattctga tgtgtatatc 540
acagacaaaa ctgtgctaga catgaggtct atggacttca agagcaacag tgctgtggcc 600
tggagcaaca aatctgactt tgcatgtgca aacgccttca acaacagcat tattccagaa 660
gacaccttct tccccagccc agaaagttcc tgtgatgtca agctggtcga gaaaagcttt 720
gaaacagata cgaacctaaa ctttcaaaac ctgtcagtga ttgggttccg aatcctcctc 780
ctgaaagtgg ccgggtttaa tctgctcatg acgctgcggc tgtggtccag ctga 834
<210> 66
<211> 924
<212> DNA
<213> Human
<400> 66
atgggaatca ggctcctctg tcgtgtggcc ttttgtttcc tggctgtagg cctcgtagat 60
gtgaaagtaa cccagagctc gagatatcta gtcaaaagga cgggagagaa agtttttctg 120
gaatgtgtcc aggatatgga ccatgaaaat atgttctggt atcgacaaga cccaggtctg 180
gggctacggc tgatctattt ctcatatgat gttaaaatga aagaaaaagg agatattcct 240
gaggggtaca gtgtctctag agagaagaag gagcgcttct ccctgattct ggagtccgcc 300
agcaccaacc agacatctat gtacctctgt gccagcagta accagatcta tggctacacc 360
ttcggttcgg ggaccaggtt aaccgttgta gaggacctga acaaggtgtt cccacccgag 420
gtcgctgtgt ttgagccatc agaagcagag atctcccaca cccaaaaggc cacactggtg 480
tgcctggcca caggcttctt ccctgaccac gtggagctga gctggtgggt gaatgggaag 540
gaggtgcaca gtggggtcag cacggacccg cagcccctca aggagcagcc cgccctcaat 600
gactccagat actgcctgag cagccgcctg agggtctcgg ccaccttctg gcagaacccc 660
cgcaaccact tccgctgtca agtccagttc tacgggctct cggagaatga cgagtggacc 720
caggataggg ccaaacccgt cacccagatc gtcagcgccg aggcctgggg tagagcagac 780
tgtggcttta cctcggtgtc ctaccagcaa ggggtcctgt ctgccaccat cctctatgag 840
atcctgctag ggaaggccac cctgtatgct gtgctggtca gcgcccttgt gttgatggcc 900
atggtcaaga gaaaggattt ctga 924
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Т-КЛЕТОЧНЫЕ РЕЦЕПТОРЫ | 2017 |
|
RU2775623C2 |
| Т-КЛЕТОЧНЫЕ РЕЦЕПТОРЫ | 2018 |
|
RU2762255C2 |
| КОНСТРУКТЫ Т-КЛЕТОЧНОГО РЕЦЕПТОРА И ИХ ПРИМЕНЕНИЕ | 2019 |
|
RU2785954C2 |
| Т-КЛЕТОЧНЫЕ РЕЦЕПТОРЫ, СПЕЦИФИЧНЫЕ В ОТНОШЕНИИ КОМПЛЕКСА ОПУХОЛЕВЫЙ АНТИГЕН NY-ESO-1/HLA-A*02 | 2016 |
|
RU2775394C2 |
| ХИМЕРНЫЕ КОНСТРУКЦИИ АНТИТЕЛО/Т-КЛЕТОЧНЫЙ РЕЦЕПТОР И ИХ ПРИМЕНЕНИЯ | 2016 |
|
RU2767209C2 |
| ВЫСОКОАФФИННЫЙ Т-КЛЕТОЧНЫЙ РЕЦЕПТОР ДЛЯ РАСПОЗНАВАНИЯ АНТИГЕНА AFP | 2019 |
|
RU2836365C2 |
| T-КЛЕТОЧНЫЕ РЕЦЕПТОРЫ, РАСПОЗНАЮЩИЕ МУТАЦИЮ R175H ИЛИ Y220C В P53 | 2020 |
|
RU2830061C2 |
| УЛУЧШЕННЫЙ СИНТЕТИЧЕСКИЙ Т-КЛЕТОЧНЫЙ РЕЦЕПТОР И АНТИГЕННЫЙ РЕЦЕПТОР | 2021 |
|
RU2820497C1 |
| КОМПОЗИЦИИ Т-КЛЕТОК С НЕДОСТАТОЧНОСТЬЮ РЕЦЕПТОРОВ Т-КЛЕТОК | 2013 |
|
RU2781979C2 |
| ХИМЕРНЫЙ АНТИГЕННЫЙ РЕЦЕПТОР (CAR) С АНТИГЕНСВЯЗЫВАЮЩИМИ ДОМЕНАМИ К КОНСТАНТНОЙ ОБЛАСТИ β Т-КЛЕТОЧНОГО РЕЦЕПТОРА | 2015 |
|
RU2830064C2 |






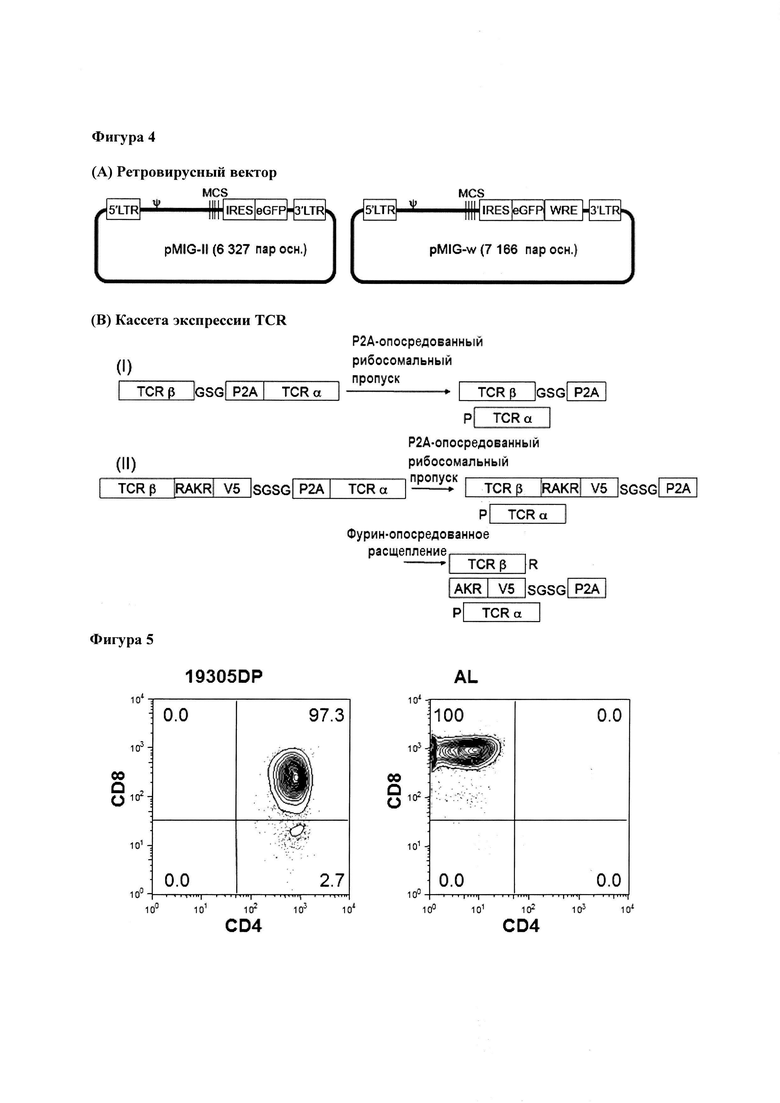





Группа изобретений относится к иммунной терапии. Предложен рекомбинантный полинуклеотид, кодирующий альфа-цепь и бета-цепь Т-клеточного рецептора (TCR), причем альфа-цепь содержит последовательность SEQ ID NO: 1, и бета-цепь содержит последовательность SEQ ID NO: 2. Предложен вектор экспрессии, кодирующий альфа-цепь и бета-цепь Т-клеточного рецептора (TCR), причем альфа-цепь содержит последовательность SEQ ID NO: 1, и бета-цепь содержит последовательность SEQ ID NO: 2. Предложены также комбинация двух вышеуказанных рекомбинантных полинуклеотидов и комбинация двух вышеуказанных векторов экспрессии. Предложен также способ профилактики и/или терапии индивидуума, у которого диагностирован, подозревается наличие или существует риск развития или рецидива рака, включающий введение индивидууму модифицированных Т-клеток человека. При этом рак включает раковые клетки, которые экспрессируют NY-ESO-1/LAGE-1 антиген. При этом Т-клетки человека содержат a) один вектор экспрессии, содержащий рекомбинантный полинуклеотид, кодирующий Т-клеточный рецептор (TCR), содержащий альфа-цепь, содержащую последовательность SEQ ID NO: 1, и бета-цепь, содержащую последовательность SEQ ID NO: 2; или b) два вектора экспрессии, причем один вектор экспрессии кодирует альфа-цепь, содержащую последовательность SEQ ID NO: 1, а второй вектор экспрессии кодирует бета-цепь, содержащую последовательность SEQ ID NO: 2. Группа изобретений может эффективно использоваться в адаптивной иммунной терапии. 5 н. и 7 з.п. ф-лы, 10 ил., 1 табл., 2 пр.
1. Рекомбинантный полинуклеотид, который кодирует альфа-цепь и бета-цепь Т-клеточного рецептора (TCR), причем альфа-цепь содержит последовательность SEQ ID NO: 1, и бета-цепь содержит последовательность SEQ ID NO: 2.
2. Комбинация двух полинуклеотидов, которые кодируют альфа-цепь и бета-цепь Т-клеточного рецептора (TCR), причем альфа-цепь содержит последовательность SEQ ID NO: 1, и бета-цепь содержит последовательность SEQ ID NO: 2.
3. Вектор экспрессии, который кодирует альфа-цепь и бета-цепь Т-клеточного рецептора (TCR), причем альфа-цепь содержит последовательность SEQ ID NO: 1, и бета-цепь содержит последовательность SEQ ID NO: 2.
4. Вектор экспрессии по п. 3, причем вектор экспрессии присутствует в клетке.
5. Вектор экспрессии по п. 4, причем вектор экспрессии присутствует в клетке, которая представляет собой CD4+ Т-клетку или CD8+ Т-клетку.
6. Комбинация двух векторов экспрессии, которые кодируют альфа-цепь и бета-цепь Т-клеточного рецептора (TCR), причем альфа-цепь содержит последовательность SEQ ID NO: 1, и бета-цепь содержит последовательность SEQ ID NO: 2.
7. Комбинация двух векторов экспрессии по п. 6, причем указанная комбинация присутствует в клетке.
8. Комбинация двух векторов экспрессии по п. 7, причем указанная комбинация присутствует в клетке, которая представляет собой CD4+ Т-клетку или CD8+ Т-клетку.
9. Способ профилактики и/или терапии индивидуума, у которого диагностирован, подозревается наличие или существует риск развития или рецидива рака, при этом рак включает раковые клетки, которые экспрессируют NY-ESO-1/LAGE-1 антиген, при этом способ включает введение индивидууму модифицированных Т-клеток человека, содержащих:
a) один вектор экспрессии, содержащий рекомбинантный полинуклеотид, кодирующий Т-клеточный рецептор (TCR), содержащий альфа-цепь, содержащую последовательность SEQ ID NO: 1, и бета-цепь, содержащую последовательность SEQ ID NO: 2; или
b) два вектора экспрессии, причем один вектор экспрессии кодирует альфа-цепь, содержащую последовательность SEQ ID NO: 1, а второй вектор экспрессии кодирует бета-цепь, содержащую последовательность SEQ ID NO: 2.
10. Способ по п. 9, в котором раковые клетки выбирают из рака мочевого пузыря, клеток рака головного мозга, клеток рака молочной железы, клеток рака желудка, клеток рака пищевода, клеток рака головы и шеи, гепатобилиарных раковых клеток, клеток рака почки, клеток рака яичника, клеток немелкоклеточного рака легкого, клеток миеломы, клеток рака предстательной железы, клеток саркомы, клеток рака яичка, клеток меланомы или их комбинации.
11. Способ по п. 9 или 10, включающий извлечение Т-клеток из индивидуума перед введением и модификацию Т-клеток путем введения в Т-клетки вектора экспрессии.
12. Способ по п. 11, в котором один вектор экспрессии кодирует альфа-цепь, содержащую последовательность SEQ ID NO:1, и бета-цепь, содержащую последовательность SEQ ID NO: 2.
| ZHAO, YANGBING, et al | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| DATABASE, UniProtKB, A0A0B4J275 (TVA17_HUMAN), 04.03.2015 [найдено 18.08.2020] | |||
| Найдено онлайн: URL: | |||
Авторы
Даты
2021-07-29—Публикация
2017-01-06—Подача