Область техники.
Настоящее изобретение относится к системе и способу интеграции секвенирования полинуклеотидов и анализа фенотипов биологических частиц посредством детерминированного штрихкодирования. В высокопроизводительном анализе система и способ сохраняют связи между фенотипом биологической частицы, оцениваемым неразрушающими методами (например, оптически), и данными секвенирования полинуклеотидов, содержащихся в биологической частице.
Настоящая заявка на патент закрепляет приоритет по предварительной патентной заявке США № 62/562, 115, озаглавленной «Поточный анализ с детерминированным штрихкодированием», поданной 22 сентября 2017 г., и по предварительной заявки США № 62 / 577,905, озаглавленной «Объединение высокопроизводительного секвенирования отдельных частиц с другими методами анализа отдельных частиц посредством детерминированного штрихкодирования», поданной 27 октября 2017 года. Полное описание указанных предварительных заявок доступно по ссылкам, приведенным в данной заявке.
Определение терминов.
BP – биологическая частица, содержащая генетический материал (например, клетка или органелла, такая как митохондрия).
Фенотип – совокупность информации, касающейся функциональных свойств, молекулярного состава, структуры и морфологии отдельной BP. Например, фенотип может быть изучен с помощью оптического анализа в потоке или анализа изображения.
FC – проточная цитометрия.
FACS – Сортировка клеток с флуоресцентной активацией или, в широком смысле, любая сортировка BP на основе FC. Геном/транскриптом – все полинуклеотидные (ДНК/РНК) последовательности в данной BP.
mSCS – массовое (или высокопроизводительное) секвенирование BP, индивидуальных клеток или иных BP, не только клеток. mSCS определят геном/транскриптом каждой BP в больших совокупностях, чаще всего посредством штрихкодирования полинуклеотидного содержимого каждой BP с последующим секвенированием совокупного кодированного генетического материала множества BP. Штрихкодирование полинуклеотидных молекул из каждой BP осуществляется с использованием олигонуклеотидных меток с уникальными последовательностями. Чаще всего в системах штрихкодирования используется поток микрочастиц с прикрепленными кодирующими метками (олигонуклеотидами) в чипе, и каждая микрочастица несёт случайную, но уникальную последовательность для штрихкодирования. Каждая анализируемая BP совмещается с одной микрочастицей в микрокапле, где выполняется штрихкодирование. Затем микрокапли объединяются и секвенируются совместно. После секвенирования совмещенных полинуклеотидов из множества BP, кодирующие последовательности позволяют идентифицировать исходные полинуклеотидные последовательности, происходящие из одной и той же микрочастицы.
IDBC - Идентифицируемый детерминировано кодирующий носитель (кодирующих олигонуклеотидов) для детерминированного штрихкодирования, который состоит из идентифицируемого ядра и олигонуклеотидов заранее определенной последовательности, прикрепленных к данному ядру. «Идентифицируемый» указывает на способность каждого конкретного носителя быть идентифицированным (ID), например, с помощью уникальной комбинации флуорофоров, или с помощью RFID, или по его положению на чипе или по месту в последовательности потока.
GIDBC - генератор идентифицируемых детерминировано кодирующих носителей (кодирующих олигонуклеотидов) для детерминированного штрихкодирования.
LIF/LS - лазерно-индуцированная флуоресценция / светорассеивание для проточного анализа.
FISH - флуоресцентная цитологическая гибридизация in situ.
Предшествующий уровень техники.
Влияние технологии на развитие биомедицинских исследований. С быстрым развитием методов биоанализа и обработки данных изучение усреднённых по совокупности свойств клеток и органелл все более устаревает, и его заменяет высокопроизводительный анализ отдельных BP. Такой широкомасштабный сдвиг в биомедицинских исследованиях обусловлен увеличением объема биологических данных, подчеркивающих значимость гетерогенности частиц BP (например, гетерогенности клеток в данной популяции). В настоящее время mSCS обеспечивает беспрецедентное понимание клеточной гетерогенности в экспрессии генов, манифестации генотипа в фенотипе, степени повреждения ДНК, регуляторных мутаций и т. д. Эта генетическая гетерогенность включает как клетки в целом так и их органеллы, например, митохондрии. В ответ на экологические, биологические и клеточные факторы генетическая гетерогенность BP трансформируется в фенотипическую гетерогенность. Параметры фенотипа клеток можно измерять несколькими способами, но FC является одним из самых высокопроизводительных методов оценки параметров клеточных фенотипов (например, функциональных свойств и морфологии). Возможность одновременно определять фенотип и геном/транскриптом каждой BP в большой популяции могла бы кардинально изменить парадигму биомедицинских исследований.
Настоящее изобретение полезно для анализа BP, включая, но не ограничиваясь такими BP как: соматические клетки хоста, трансформированные клетки хоста, патогенная биота, симбиотическая биота, митохондрии (особенно в ооцитах), сперматозоиды, тромбоциты (в анализе содержания митохондрий), ядра, ядрышки, рибосомы, вирусы и экзосомы.
В биомедицинских исследованиях отсутствует и срочно требуется инструмент или высокопроизводительная технологическая платформа, которая может однозначно сопоставить информацию mSCS для конкретной клетки с параметрами фенотипа той же самой клетки. Случайность последовательностей кодирующих олигонуклеотидов в существующих платформах mSCS не обеспечивает идентификации конкретной BP, содержащей полинуклеотиды, она только устанавливает, происходят ли полинуклеотиды из одной и той же BP или нет. Это не позволяет соотнести измеренные FC фенотипические свойства с геномом/транскриптомом отдельных BP. Возможно, самой близкой технологией, которая действительно сопоставляет некоторые фенотипические свойства с транскриптомами отдельных BP, является недавно опубликованный подход, названный CITE-SEQ, где мечение комплексом антитело-олигонуклеотид дает возможность ограниченной идентификации фенотипа с использованием маркеров клеточной поверхности; тем не менее, эта платформа не обеспечивает исчерпывающее высокопроизводительное сопоставление данных анализа генома / транскриптома и фенотипа индивидуальных клеток. Настоящее изобретение преодолевает эти ограничения существующих методологий mSCS, доступных биомедицинскому исследовательскому сообществу.
Новые возможности и применимость. Точное определение генома/ транскриптома и соответствующего фенотипа для каждой BP в гетерогенной популяции необходимо для более глубокого понимания многих биомедицинских проблем. Примеры возможностей, которые предоставит данное изобретение, включают: анализ развития рака, в частности формирование поликлональности при метастатическом раке, определение взаимосвязей между стволовыми клетками и канцерогенезом, понимание механизмов развития резистентности к терапии и манифестации генотипа в фенотипе, исследования взаимодействий отдельных вирусов/бактерий/патогенов с отдельными клетками, исследования сложных микробных сообществ и их взаимодействий с хостом, определение роли митохондриальной гетероплазмидности в компрометации апоптотической антипролиферации и т. д. В следующих параграфах мы кратко остановимся на некоторых из этих биомедицинских применений, чтобы предоставить выборку из широких возможностей этой технологии:
Определение взаимосвязей между стволовыми клетками и канцерогенезом. Терапия стволовыми клетками эффективна для лечения определенных патологий, но частично ограничена потенциальной канцерогенностью. Популяция стволовых клеток всегда является гетерогенной из-за повреждений ДНК, мутационной нагрузки и изменчивости транскриптома, вызванной окружающей средой. Эта неоднородность проявляется в фенотипических вариациях, иногда канцерогенных. Следовательно, необходим глубокий анализ проявлений генома и транскриптома в фенотипе для больших популяций.
Исследования канцерогенеза. Канцерогенез, как правило, является стохастическим процессом, когда комбинация генетических изменений (часто опосредованных ретровирусами, транспозонами, плазмидами, вирусными факторами) и регуляторного влияния (изменение транскриптома) приводит к злокачественному фенотипу. Очень важно выявить связи генотип-транскриптом-фенотип на уровне индивидуальных клеток в больших клеточных сообществах.
Исследования поликлонального рака. Раковые клетки часто имеют повышенную частоту мутаций и, как следствие, повышенную приспособляемость. Мутагенная природа большинства методов лечения рака еще больше повышает адаптивность. Раннее обнаружение клонов, устойчивых к лекарствам и иммунотерапии, имеет важное значение, особенно для рассеянных раковых клеток. Обнаружение клеток с необычным метаболизмом, апоптотической сигнатурой или признаками старения в сочетании с непосредственным определением соответствующих мутаций в геноме/транскриптоме изучаемых клеток поможет идентифицировать злокачественные клоны и, следовательно, модифицировать терапию.
Исследования клеточно-вирусных взаимодействий. Взаимодействие клеток с вирусными частицами, в частности с онковирусами, представляет собой очень гетерогенный процесс, при котором только небольшая часть клеток может быть восприимчивой к инфекции в зависимости от клеточного статуса и транскриптома. Обнаружение клеточно-вирусных взаимодействий и последующих фенотипических изменений с непосредственным определением соответствующего транскриптома, и судьбы вирусного генетического материала для каждой клетки будет иметь важное значение для исследований вирулентности.
Динамика бактериальных сообществ. Бактериальные сообщества, включая микробиом человека, крайне гетерогенны. Реакция микробиомов на изменяющуюся среду, такую как доступность белков, жиров, устойчивых к пищеварению и усваиваемых углеводов, присутствие токсинов, терапевтических агентов или введение новых, возможно, патогенных видов биоты чрезвычайно сложна. Бактерии могут адаптировать свой фенотип посредством индивидуальной или коллективной реакции, выделяя определенные организмы для самоубийственного высвобождения биоцидов или проявляя регуляторные мутации. В настоящее время нет доступных технологий для глубокого изучения этих сложных и специфичных изменений в индивидуальных клетках без направленного измерения функциональных свойств, морфологии, биомолекулярного содержания, транскиптома и генома каждой бактерии в обширном сообществе.
Изучение роли гетероплазмии митохондрий в нарушении механизмов апоптоза/сенессенции/митогенеза. Исследования гетероплазмии митохондрий могут определить связи между мутациями мтДНК и их повреждением, транскрипцией, функциональностью и содержанием биомолекул в митохондрии. Изучение того, как клетка маркирует отдельные митохондрии для митофагии, репликации или инициации апоптотических процессов, важно для понимания противораковых механизмов и их прогрессирующей деградации в течение жизни, а также для связанных со старением митохондриально-обусловленной нервной и мышечной дегенерации, бесплодия и наследственных патологий.
Таким образом, из уровня техники следует, что требуется система и способ, которые позволят однозначно сопоставить информацию mSCS для конкретной клетки с параметрами фенотипа той же самой клетки, соотнести измеренные FC фенотипические свойства с геномом/транскриптомом отдельных BP, с высокой производительностью.
Настоящее изобретение относится к системе и способу интеграции секвенирования полинуклеотидов и анализа фенотипов биологических частиц посредством детерминированного штрихкодирования. В высокопроизводительном анализе система и способ сохраняют связи между фенотипом биологической частицы, оцениваемым неразрушающими способами (например, оптически), и данными секвенирования полинуклеотидов, содержащихся в биологической частице.
Технический результат заключается в обеспечении возможности точного определение неразрушающим способом генома/ транскриптома и соответствующего фенотипа для каждой BP в гетерогенной популяции. Система и способ обеспечивают исчерпывающее высокопроизводительное сопоставление данных анализа генома / транскриптома и фенотипа индивидуальных клеток.
Технический результат достигается тем, что система для анализа фенотипа и последовательности полинуклеотидов биологической частицы, содержит:
- анализатор фенотипа, где указанный анализатор фенотипа предназначен для определения фенотипа указанной биологической частицы;
- генератор идентифицируемого олигонуклеотидного кодирующего носителя, где кодирующий олигонуклеотид с заранее известной последовательностью, прикреплен к указанному носителю, используется индивидуальный носитель для каждой биологической частицы и применяется уникальный и предварительно определенный штрихкод для каждого полинуклеотида каждой биологической частицы;
- микрореактор, выполненный с возможностью приема указанной биологической частицы из указанного анализатора фенотипа и приема указанного носителя кодирующих олигонуклеотидов из указанного генератора;
- секвенатор указанной предварительно определенной последовательности указанного кодирующего олигонукеголеотида и указанной последовательности полинуклеотидов указанной биологической частицы после того, как указанный кодирующий олигонуклеотид лигируют с указанными полинуклеотидами указанной биологической частицы
В отличие от существующих способов mSCS с частицами, несущими уникальные, но случайные кодирующие олигонуклеотиды, в подходе детерминированного штрихкодирования настоящего изобретения используется IDBC для каждой BP и применяется уникальный и предварительно определенный штрихкод для каждого полинуклеотида каждой BP.
Указанная биологическая частица и указанный носитель кодирующих олигонуклеотидов инкапсулированы в капле в указанном микрореакторе.
Предусмотрено, что анализатор фенотипа представляет собой проточный цитометр.
Предусмотрено, что анализатор фенотипа представляет собой микроскоп.
Предусмотрено, что носитель представляет собой магнитную частицу.
Предусмотрено, что носитель представляет собой микроячейку на поверхности чипа.
Предусмотрено, что система содержит аккумулятор биологических частиц между указанным анализатором фенотипа и указанным микрореактором.
Технический результат достигается тем, что способ анализа фенотипа и последовательности полинуклеотидов биологической частицы, включает стадии:
оптического анализа указанной биологической частицы для определения фенотипа указанной биологической частицы;
записи указанного фенотипа указанной биологической частицы;
генерирования носителя кодирующих олигонуклеотидов, где кодирующий олигонуклеотид предварительно определенной последовательности прикреплен к указанному носителю; используется индивидуальный носитель для каждой биологической частицы и применяется уникальный и предварительно определенный штрихкод для каждого полинуклеотида каждой биологической частицы;
помещение указанной биологической частицы и указанного носителя кодирующих олигонуклеотидов в микрореактор;
лизис указанной биологической частицы для высвобождения указанных полинуклеотидов указанной биологической частицы;
лигирование указанных кодирующих олигонуклеотид ов с указанными полинуклеотидами указанной биологической частицы для образования генетического комплекса;
секвенирование указанного генетического комплекса для определения указанной последовательности указанных полинуклеотидов указанной биологической частицы;
сопоставление указанного фенотипа указанной биологической частицы с указанной последовательностью указанных полинуклеотидов указанной биологической частицы.
Предусмотрено, что указанная биологическая частица и указанный носитель кодирующих олигонуклеотидов инкапсулированы в капле в указанном микрореакторе.
Предусмотрено, что проточный цитометр выполняет этап оптического анализа указанной биологической частицы для определения фенотипа указанной биологической частицы.
Предусмотрено, что указанная биологическая частица подается в аккумулятор биологических частиц до того, как указанная биологическая частица помещается в указанный микрореактор.
Предусмотрено, что способ содержит этап анализа исходящего из указанного микрореактора на наличие ошибок.
Предусмотрено, что указанная стадия генерирования носителя кодирующих олигонуклеотидов включает выращивание олигонуклеотида на частице.
Предусмотрено, что стадия выращивания олигонуклеотида на частице включает фотоактивацию.
Предусмотрено, что включает стадию иммобилизации указанной частицы в ловушке на чипе.
Предусмотрено, что стадия генерирования носителя кодирующих олигонуклеотидов включает выращивание олигонуклеотида на чипе.
Техническое решение поясняется графическими материалами, на которых изображено:
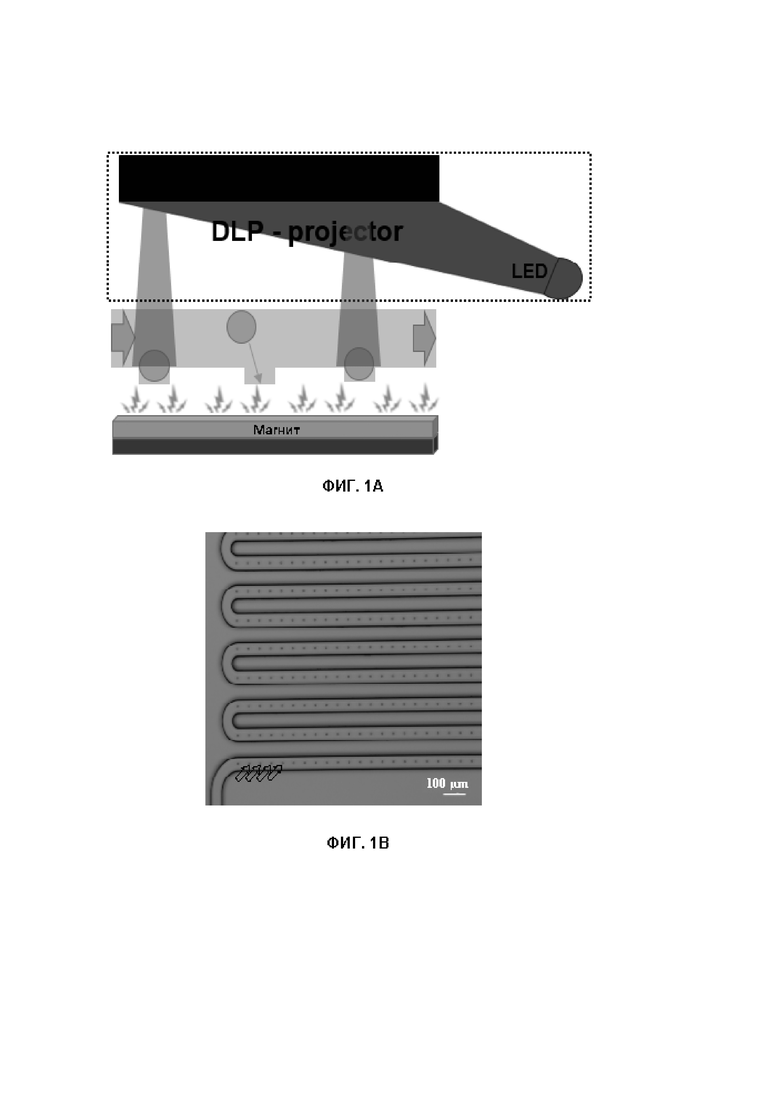
Фиг. 1А показана схема GIDBC. Конкретный вариант осуществления изобретения на Фиг. 1A использует идентификацию IDBC по положению, обратимую иммобилизацию магнитным полем и фотоактивацию для выращивания олигонуклеотидов. Магнитные частицы (круги) показаны обратимо захваченными в углублениях поверхности чипа внешним магнитным полем (искры). Захваченные частицы индивидуально освещают для снятия фотоудаляемой защиты и затем присоединяют к ним нуклеотиды. Обозначено: DLP-Projector – DLP Проектор.
Фиг.1В показан вид сверху на SU-8 мастер-модель прототипа чипа с извилистым микрофлюидным каналом в области фотообработки и магнитными ловушками в канале, отмеченными стрелками.
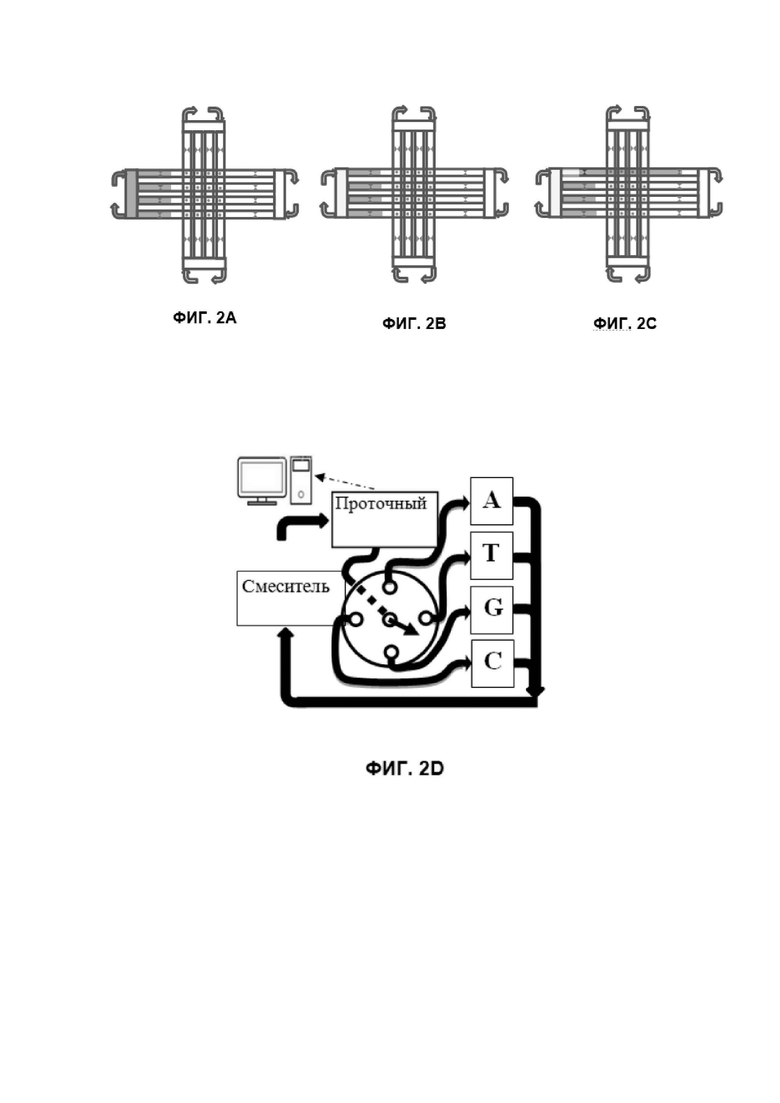
Фиг.2А-2С показаны схемы элемента чипа с перекрестным потоком для синтеза олигонуклеотидов. На Фиг. 2А показано, как реагент вводится в левый резервуар и левую область/ левые оконечности каналов. На Фиг.2В показано, как резервуар заполнен изолирующей средой для разрушения электрического контакта между каналами. Фиг. 2C показывает электроосмотическое перемещение реагента в рабочую область после приложения электрического потенциала к электродам верхнего канала. Верхний горизонтальный канал в чипе можно использовать для добавления нуклеотида ко всем пересечениям в верхнем ряду, а затем для снятия защиты с якорей для выращивания олигонуклеотидов на этих пересечениях, чтобы впоследствии добавить нуклеотид к некоторым из них через вертикальные каналы.
Фиг.2D - схема, иллюстрирующая синтез отслеживаемого смешения. Проточный детектор используется для отслеживания каждого IDBC в каждом раунде присоединения нуклеотидов. Четырехходовой клапан направляет по одной четверти суспензии IDBC в камеры для присоединения нуклеотидов A, T, G, C, соответственно. После присоединения нуклеотидов A, T, G и C, IDBC возвращаются в смеситель из всех четырех камер и готовы к следующему этапу выращивания кодирующих олигонуклеотидов. В результате смесь IDBC с уникальными штрихкодами дополняется базой данных, где каждый IDBC-идентификатор связан с определенной кодирующей последовательностью.
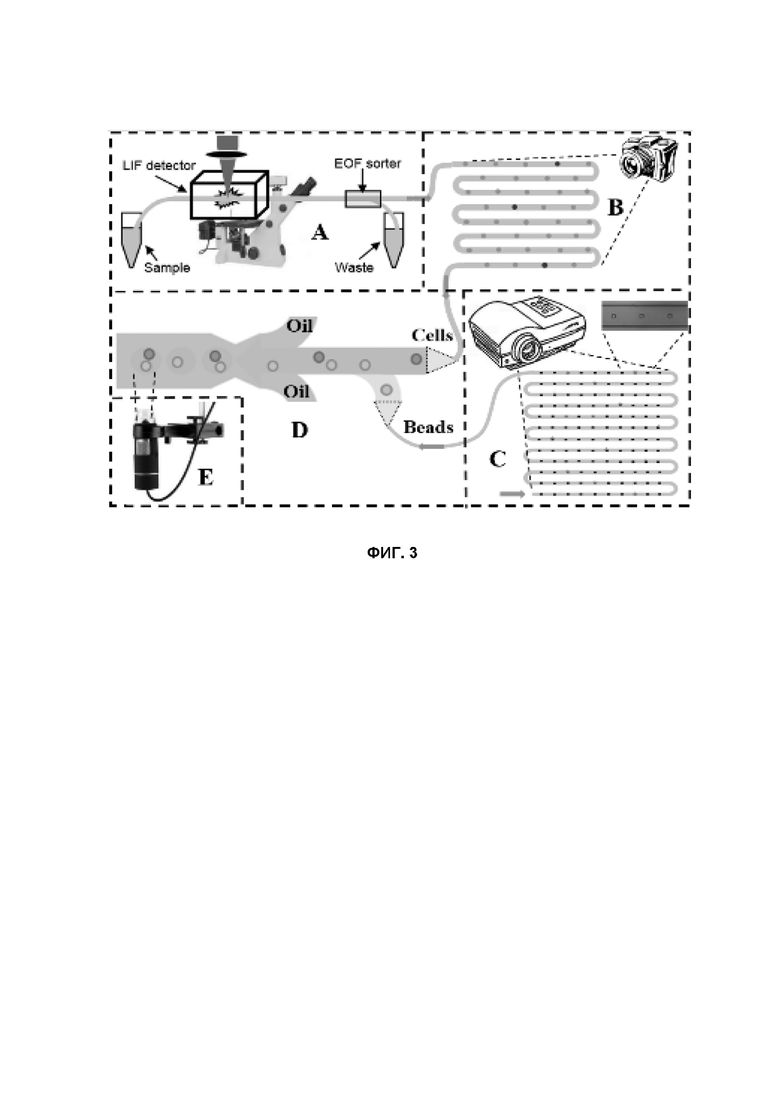
Фиг.3 представляет собой схему, показывающую компоненты настоящего изобретения в пяти блоках. Блок A представляет собой цитометр (FC) или устройство для сортировки клеток с флуоресцентной активацией (FACS), Блок B представляет собой аккумулятор клеток с камерой, анализирующей положения контрольных микросфер (самые темные три круга), Блок C представляет собой GIDBC с IDBC (например, микросферы), удерживаемые в отдельных ловушках, в то время как олигонуклеотиды с предварительно определенной последовательностью выращиваются путем селективной фотоактивации с использованием DLP-проектора, блок D является генератором капель, в которых происходит кодирование, а блок E является детектором синхронизации. Обозначено: LIF-detector- LIF-детектор, EOF-sorter - EOF-сортировщик, Sample - Образец, Waste – Отходы. Oil - Масло Cells - Клетки, Beads- Микросферы.
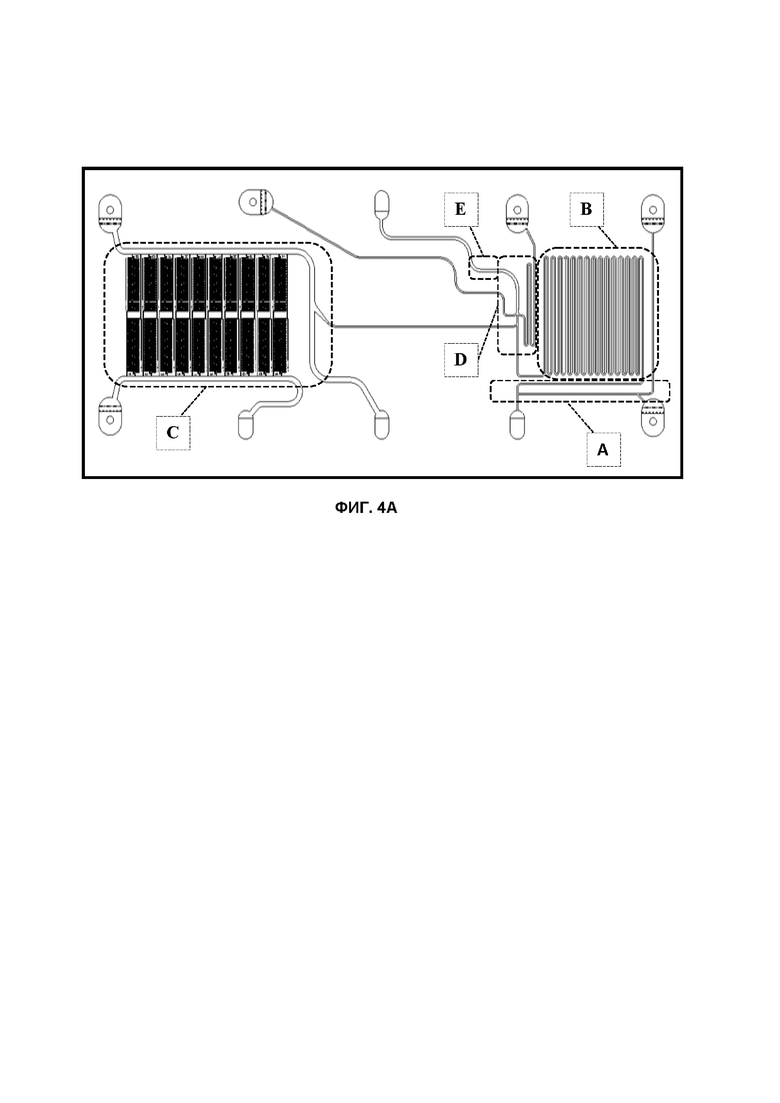
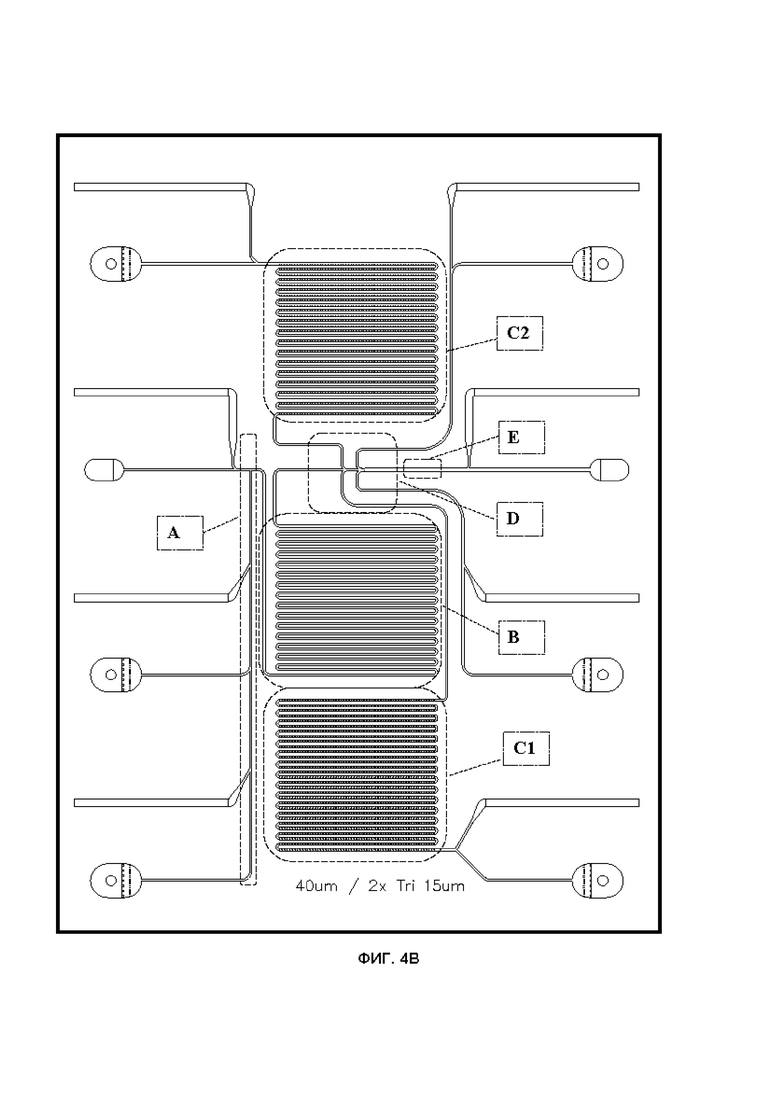
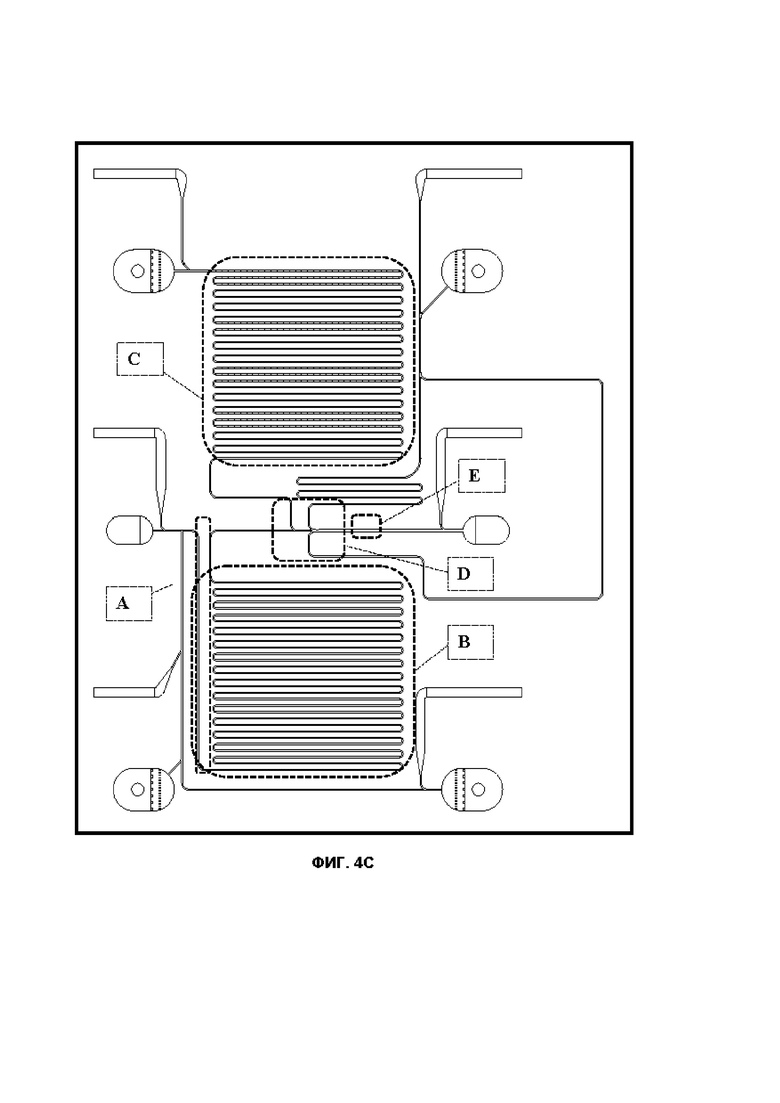
Фиг. 4А-4С показывают примеры компоновки интегрированных чипов. На Фиг. 4А показана компоновка чипа, содержащая все компоненты прибора с гидродинамическими ловушками. Фиг. 4B-4C показывают компоновки чипа, содержащие все компоненты приборов с магнитными ловушками. На Фиг.4В показана компоновка с минимизированными разъёмами, а на Фиг.4С показана компоновка с двойным GIDBC и не включающую петлю в морфологию.
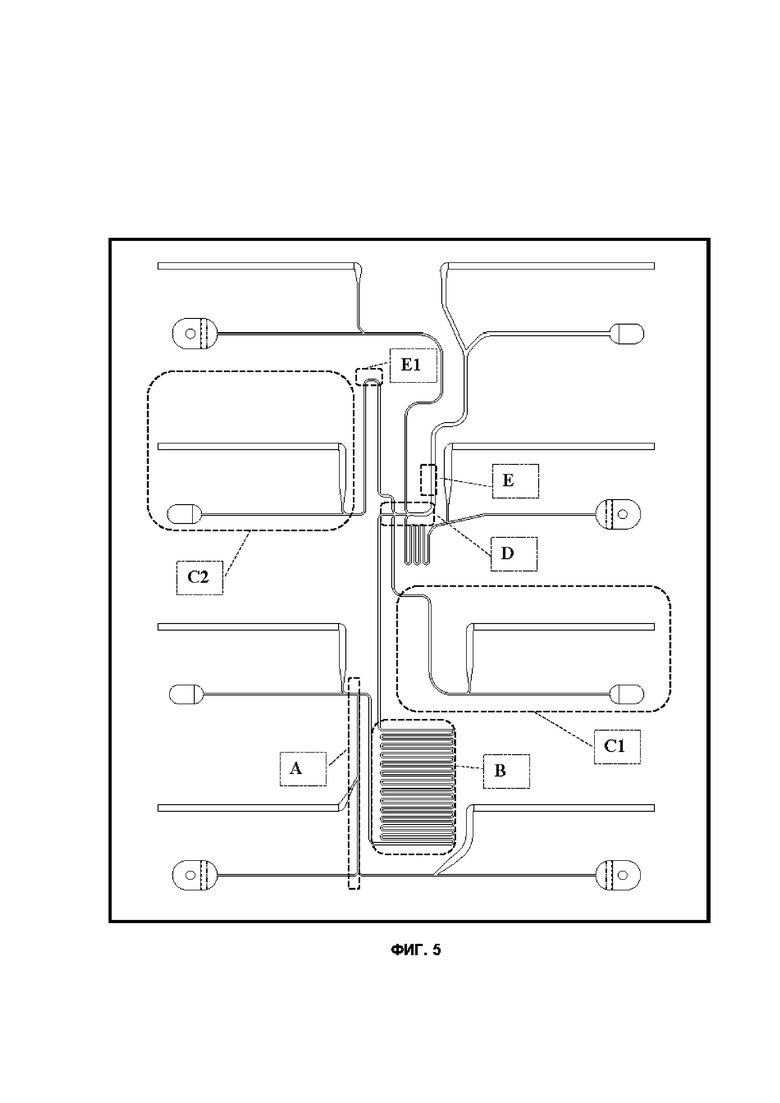
Фиг. 5 приведен пример схемы чипа со всеми интегрированными компонентами, кроме GIDBC. Компоновка чипа включает в себя выделенную область проточного обнаружения IDBC для синхронизации (E1).
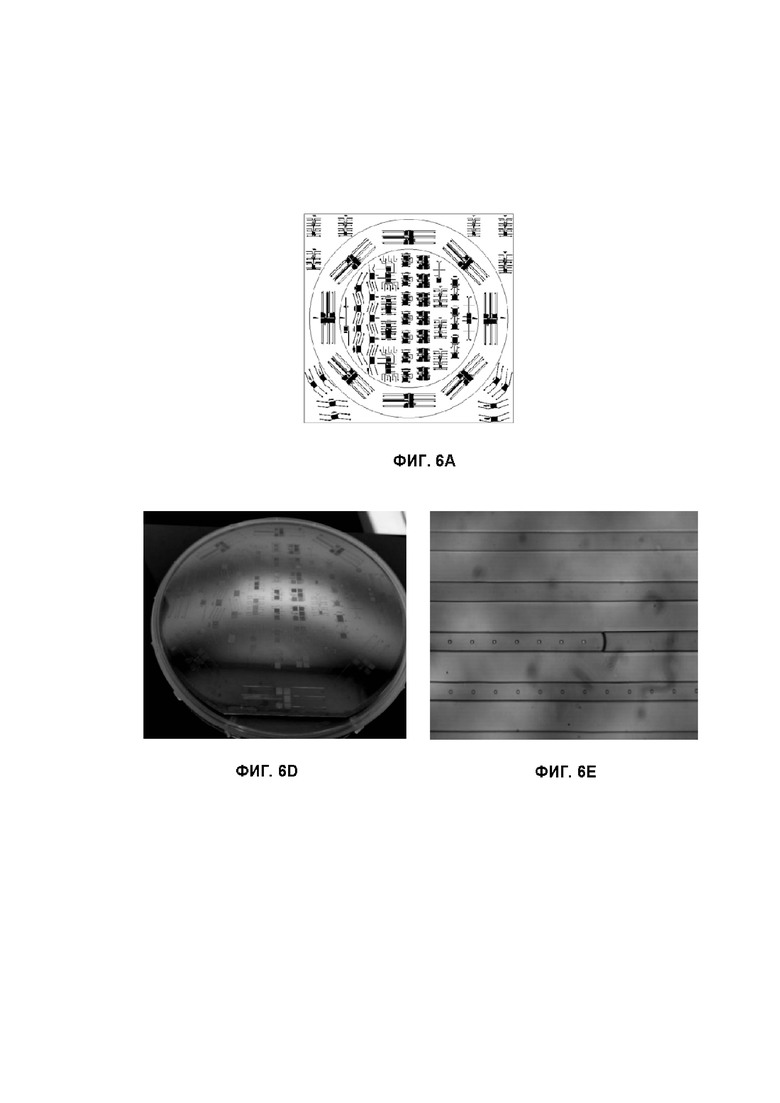
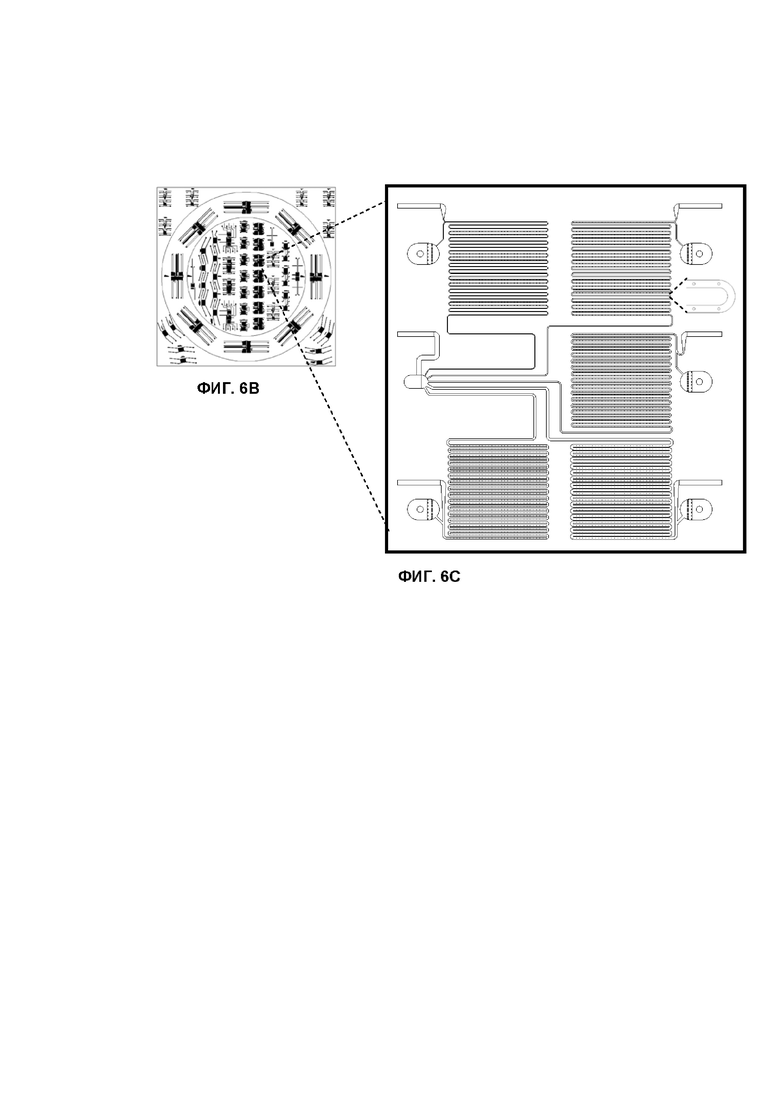
Фиг. 6А-6Е изображены компоновки для двухслойных чипов PDMS. На Фиг.6А показана маска для слоя толщиной 5 мкм, содержащего ловушки. На Фиг.6В показана маска для слоя толщиной 40 мкм, содержащего каналы. Эта компоновка подходит для изготовления двухслойных мастеров 105 мм (внутренний круг) и 130 мм (внешний круг) с дополнительными чипами по углам. Она содержит несколько видов чипов, включая чипы для оценки оптимальной геометрии каналов и ловушек. На Фиг.6С показан пример компоновки, содержащей пять областей тестирования (квадраты 3х3 мм, заполненные извилистым каналом с ловушками на 900 и 1800 микросфер в каждой), овальные входы с фильтрами, прямоугольные входы без фильтров и область проточной детекции, ограниченную разъёмом (в центре слева). Вверху справа вставка показывает увеличенные ловушки. На Фиг.6D показано изображение SU-8 мастер-модели, выполненной с использованием масок на Фиг.6А и Фиг.6В. На Фиг.6E показано изображение области готового чипа из PDMS/стекла, которая наполовину заполнена рабочим буфером. Ловушки видны только в заполненной воздухом половине канала из-за более высокой разницы в показателе преломления.
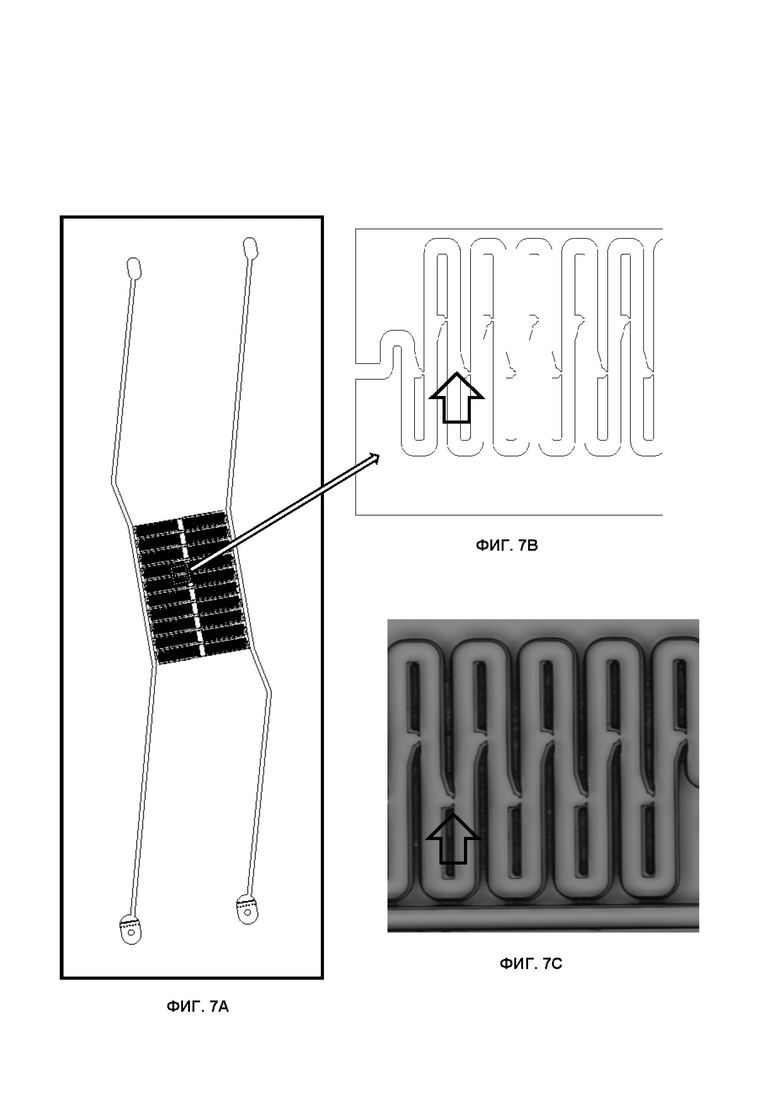
На Фиг.7А показан пример компоновки маски для изготовления чипа с гидродинамическими ловушками. Фиг.7В дает увеличенное изображение ловушек из Фиг.7А. На Фиг. 7C показан элемент SU-8 мастер-модели, выполненной с компоновкой Фиг. 7A и Фиг. 7B. Стрелка обозначает ловушку для микросферы на базе микропотока по укороченной траектории.
Фиг. 8А-8D показаны магнитные ловушки различной морфологии. Фиг. 8A-8C показывают увеличенные изображения различных комбинаций ловушек канала для вариантов компоновки чипов, показанных на Фиг. 4C, 6C и 6E. Фиг.8D показана область готового чипа из PDMS/стекла, изготовленного с использованием мастера, показанного на Фиг.6D, и наполовину заполненного рабочим буфером. Ловушки видны только в заполненной воздухом половине из-за более высокой разницы в показателе преломления.
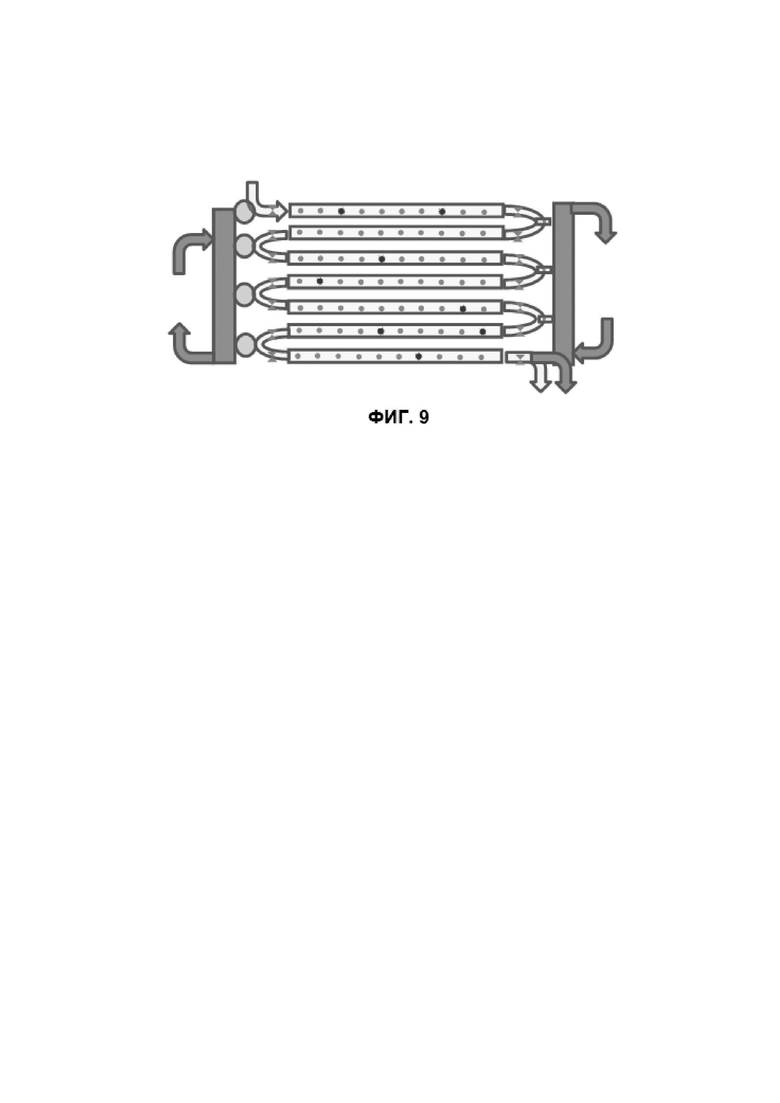
Фиг.9 показана схема варианта реализации GIDBC с независимым EOF контролем потока среды в каждом горизонтальном канале. Вертикально ориентированные боковые каналы обеспечивают параллельное промывание во время синтеза олигонуклеотидов. Электроды (двойные треугольники) обеспечивают независимую генерацию EOF в отдельных горизонтальных каналах после заполнения вертикальных каналов изолятором. Большие круги представляют резервуары электролита. Импульсная лазерная накачка может использоваться вместо EOF для избирательной прокачки среды.
Осуществление технического решения.
Настоящее изобретение относится к системе и способу интеграции секвенирования полинуклеотидов и анализа фенотипов биологических частиц посредством детерминированного штрихкодирования. В высокопроизводительном анализе система и способ сохраняют связи между фенотипом биологической частицы, оцениваемым неразрушающими методами (например, оптически), и данными секвенирования полинуклеотидов, содержащихся в биологической частице.
Точнее говоря, настоящее изобретение направлено на создание системы анализа фенотипа и полинуклеотидных последовательностей биологической частицы, включающей анализатор фенотипа, где анализатор фенотипа предназначен для определения фенотипа биологической частицы; генератор идентифицируемых, детерминированно кодирующих носителей, где кодирующий олигонуклеотид заранее определенной последовательности прикреплен к идентифицируемому ядру кодирующих носителей; микрореактор, сконфигурированный для приема биологической частицы из анализатора фенотипа и идентифицируемого кодирующего носителя из генератора, для высвобождения полинуклеотидов из биологической частицы, для выполнения обратной транскрипции, если это необходимо для анализа РНК, для присоединения кодирующих олигонуклеотидов из носителя к полинуклеотидам из биологической частицы, и для амплификации получающихся в результате кодированных полинуклеотидов; и секвенатор для получения последовательностей кодированных полинуклеотидов.
Кроме того, настоящее изобретение относится к способу анализа фенотипических и полинуклеотидных последовательностей биологической частицы, включающему этапы неразрушающего анализа биологической частицы для определения фенотипа биологической частицы; записи фенотипа биологической частицы; генерации кодирующего носителя, в котором кодирующий олигонуклеотид с заранее определенной последовательностью присоединен к идентифицируемому ядру кодирующего носителя; передачи биологической частицы и идентифицируемого носителя кодирующего олигонуклеотида в микрореактор, и сопряжения кодирующего носителя с биологической частицей; лизирования биологической частицы для высвобождения полинуклеотидов биологической частицы; обратной транскрипции РНК с биологической частицы (при необходимости), лигирования кодирующего олигонуклеотида с полинуклеотидами биологической частицы для образования кодированных полинуклеотидов; секвенирования кодированных полинуклеотидов для определения последовательностей полинуклеотидов из биологической частицы; и сопоставления фенотипа биологической частицы с последовательностями полинуклеотидов биологической частицы.
Эти и другие особенности, объекты и преимущества настоящего изобретения станут более понятными из рассмотрения следующего подробного описания предпочтительных вариантов осуществления и прилагаемых формул изобретения в сочетании с рисунками, как описано ниже.
Частные примеры осуществления.
Со ссылкой на Фиг. 1А-9, можно описать варианты осуществления системы и способа по настоящему изобретению. Настоящее изобретение относится к системе и способу анализа фенотипа и секвенированию полинуклеотидов для ДНК/РНК-содержащих частиц посредством детерминированного штрихкодирования. Система и способ направлены на сохранение связей между оптически оцениваемыми отдельными фенотипами BP и геномом/транскриптомом BP в высокопроизводительном анализе. В отличие от существующих способов mSCS с частицами, несущими уникальные, но случайные кодирующие олигонуклеотиды, в подходе детерминированного штрихкодирования настоящего изобретения используется IDBC для каждой BP и применяется уникальный и предварительно определенный штрихкод для каждого полинуклеотида каждой BP. Следовательно, IDBC является ключевым элементом данного изобретения.
Для разработки IDBC авторы изобретения рассмотрели комбинации идентифицируемых ядер и процедур выращивания олигонуклеотидов с детерминированными последовательностями. В качестве идентифицируемых ядер авторы изобретения рассматривали: (1) частицы в последовательном потоке, где конкретный IDBC идентифицируется номером в последовательности, на основе его расположения в последовательном оттоке; (2) массив микролунок или микрозон на чипе, где идентичность конкретной микрозоны определяется координатами микрозоны; (3) и частицы со встроенными уникальными идентификационными метками, которые можно идентифицировать в потоке. Последний случай включает в себя частицы с: (а) несколькими оптическими метками (например, флуоресцентными метками или метками гигантского комбинационного рассеивания); и (б) частицы с радиочастотными идентификаторами (RFID).
Затем олигонуклеотиды с известными, заранее определенными последовательностями выращивают на идентифицируемых ядрах IDBC, описанных выше, для получения IDBC посредством одной из следующих процедур выращивания: (1) фотоактивация; (2) синтез в перекрещивающихся потоках; или (3) синтез с отслеживанием смешивания.
Фотоактивация является хорошо отработанной технологией для генерации олигонуклеотидов с заранее определенной последовательностью на поверхности чипа. Вкратце, защитные группы удаляются в выбранных микрозонах при освещении светом, и один нуклеотид (A, T, G или C) присоединяется к растущим олигонуклеотидам в активированных микрозонах. Этот процесс повторяется для остальных трех нуклеотидов. Затем эти четыре цикла повторяются N-раз для N-нуклеотидных длинных олигонуклеотидов, генерируя до 4N уникальных микрозон.
Фотоактивированный синтез может давать множество областей с олигонуклеотидами с микрозона-специфическими, заранее определенными последовательностями. Количество областей и, следовательно, различных последовательностей, как правило, не ограничено и зависит только от размера чипа и разрешающей способности процесса. В настоящее время при наиболее эффективном подходе к фотоактивации используется цифровая обработка света (DLP), которая позволяет сформировать миллионы независимых микрозон для уникальных последовательностей в одной обработке.
Эта технология может применяться «как есть» для роста олигонуклеотидов на микрозонах чипа (идентифицируемый вариант ядра № 2), а также с изменениями – для выращивания олигонуклеотидов на идентифицируемых микрочастицах (идентифицируемые варианты ядра № 1 и № 3). Для последнего необходима обратимая иммобилизация микрочастиц в заранее определенных микрозонах чипа, как показано на Фиг. 1А-1В, 3, 6С, 6Е и 7А-9.
Частицы могут быть обратимо иммобилизованы (или захвачены) в заранее определенных микрозонах микрофлюидного канала, которые организованных в области фотообработки на чипе так, чтобы разместить максимальное количество этих микрозон в этой области. Возможны магнитный, гидродинамический, диэлектрофорезный, химический, вакуумный и инерционный подходы к иммобилизации. При магнитном подходе намагниченные (обладающие магнитной восприимчивостью, например, суперпарамагнитные) частицы удерживаются внешним магнитным полем в поверхностных выемках или ловушках, как показано на Фиг. 1B, 3, 4B, 4C, 6C и 6E. При гидродинамическом подходе частицы удерживаются в микроканалах сокращённой траектории, с сечением меньшим, чем у микрочастиц, как показано на Фиг. 4А и 7А-С. При диэлектрофорезном подходе микрочастицы удерживаются в ловушках, заданных электродами, посредством переменного электромагнитного поля с частотой, определяемой размером частицы. При химическом подходе частицы химически иммобилизуются с использованием расщепляемых связей (например, ковалентных, ионных, гидрофобных, ван-дер-ваальсовых, водородных и т.д.), но это связывание не должно мешать синтезу олигонуклеотидов и должно быть полностью обратимым. Силу тяжести можно использовать для очень низких потоков; а центробежная сила может быть использована на чипах в вращающемся держателе. При вакуумном подходе микрочастицы удерживаются в ловушках путем внешней генерации пониженного давления в ловушках.
Из этих альтернатив магнитная иммобилизация, как показано на Фиг.1А, является предпочтительным вариантом, за которым следует гидродинамический и диэлектрофорезный подходы. Таким образом, авторы изобретения идентифицировали способ фотоактивации с магнитным, гидродинамическим или диэлектрофорезным удерживанием, используемым для позиционирования ядер IDBC, для выращивания кодирующих олигонуклеотидов на IDBC, идентифицируемых по положению в последовательном потоке, в качестве предпочтительного варианта осуществления настоящего изобретения.
Синтез перекрещивающихся потоков является альтернативой для случаев, когда фотоактивация не подходит. В этой альтернативе олигонуклеотиды выращивают на идентифицируемых ядрах в местах пересечения индивидуально контролируемых микрофлюидных потоков, как показано на Фиг. 2А-С. Предлагается использовать чип с сеткой микрофлюидных каналов, с расположением ядер на пересечениях каналов, и с электродами, размещенными в конце каждого канала. Электроды используются для генерации EOF между каждой выбранной парой электродов и селективного управления средой между ними. В качестве альтернативы, лазерная пульсация может быть использована для управления такой средой. Окончания каналов на каждой стороне чипа подключены к резервуарам. Резервуары поэтапно заполняют реагентами для прикрепления определенного защищенного нуклеотида к растущим меткам (кодирующим олигонуклеотидам), и затем изолированной средой. Сочетание чередующегося заполнения пересекающихся каналов и смены типа присоединяемого нуклеотида приводит к выращиванию олигонуклеотида уникальной и заранее известной последовательности на IDBC на каждом пересечении. Для большей гибкости получаемых последовательностей один набор каналов может быть заполнен реагентом, снимающим защиту с якоря, а другой - реагентом, присоединяющим нуклеотиды. В качестве альтернативы, среда может управляться (прокачиваться) импульсным лазером.
Синтез с отслеживанием смешивания является предпочтительным методом выращивания олигонуклеотидов на IDBC, если используются ядра с внутренним идентификатором (RFID или оптическим). Примером IDBC с внутренним идентификатором является ядро с идентификатором, созданным комбинацией 16 флуорофоров, которые одновременно обнаруживаются во многих коммерческих цитометрах и допускают 216 = 65 536 уникальных идентификаторов (для бинарной метки). Половина образца IDBC может быть модифицирована одним флуорофором, затем смешана с немодифицированной половиной, и этот цикл повторяется с другими флуорофорами. Также возможны другие средства идентификации (например, RFID, магнитная, оптическая не флуоресцентная и т.д.). Комбинации различных идентификаторов (например, флуоресцентного и RFID) дополнительно увеличивают количество уникальных идентификаторов ядра.
В существующих mSCS со случайным штрихкодированием, набор из четырех реакторов вводит один нуклеотид на каждом этапе синтеза. После каждого шага все частицы смешиваются и делятся на четыре фракции для следующего шага. Но для IDBC с встроенными в ядра идентификаторами, авторы изобретения предлагают соединить эти реакторы и смеситель с микрофлюидными линиями с проточным детектором, как показано на Фиг. 2D. В результате каждый переход IDBC между камерой смешивания и реакторами записывается, и доступна полная история движения для каждого IDBC, которая определяет последовательность кодирующих олигонуклеотидов. Частицы, которые получили одну и ту же последовательность (то есть проходили по одному и тому же пути во всех циклах синтеза), могут быть удалены из конечной смеси с использованием сортировщика, активируемого тем же проточными детектором.
IDBC, генерируемые с помощью этих ядер и процедур выращивания олигонуклеотидов, обеспечивают несколько подходов к детерминированному штрихкодированию, которые объединяют неразрушающий анализ фенотипа (например, проточный цитометр или анализ изображений под микроскопом) биологических частиц с mSCS-анализом генома или транскриптома биологических частиц. IDBC позволяет индивидуально маркировать каждую биологическую частицу после почти любого анализа фенотипа и связывать фенотип этих биологических частиц с их индивидуальными данными секвенирования. Размер популяции анализируемых биологических частиц ограничен только производительностью секвенирования, которая является быстро растущей областью. Это гарантирует, что данное изобретение будет не только полезным в современных приложениях биомедицинских исследований, но будет еще более полезным в будущих. Даже при использовании современных технологий секвенирования анализы могут включать миллионы частиц с малыми геномами/транскриптомами или небольшими целевыми генетическими элементами. Детерминированное (или известное и предварительно определенное) штрихкодирование в настоящем изобретении способно измерять любую комбинацию оптически оцениваемых фенотипических свойств, включая, но не ограничиваясь следующими: (1) мембранный потенциал, мембранную текучесть и электропермеабилизацию мембраны; (2) внутриклеточный рН, содержание ионов и кислорода; (3) общее содержание ДНК/РНК, вариабельность числа копий, анализ хромосом и сортировка; (4) экспрессия белка, модификация белка и локализация белка; (5) присутствие внутриклеточных, внутриядерных и поверхностных антигенов; (6) ферментативная активность; (7) жизнеспособность клеток, объем и морфологическая сложность; (8) наличие множественной лекарственной устойчивости; (9) адгезия (например, патогена к клетке-хосту); (10) параметры апоптоза посредством измерения деградации ДНК, потенциала митохондриальной мембраны и изменения её проницаемости; и (11) концентрация клеточных пигментов и трансгенных продуктов in vivo (например, зеленый флуоресцентный белок).
Вариант осуществления изобретения.
В одном предпочтительном варианте осуществления IDBC генерируются с использованием частиц (например, микросфер) в последовательном потоке в качестве идентифицируемых ядер с фотоактивацией в качестве процедуры для роста олигонуклеотидов на микросферах. Использование этих IDBC в системе по настоящему изобретению может установить взаимно-однозначное соответствие между информацией о последовательностях полинуклеотидов индивидуальных клеток и данными проточной цитометрии в высокопроизводительном анализе.
Как показано на Фиг. 3, FC собирает и записывает информацию, специфичную для отдельной BP, и направляет отсортированные BP в систему через дополнительный аккумулятор клеток. Параллельно микрофлюидный чип обратимо иммобилизует микрочастицы в заранее заданном массиве. Несколько стадий фотоактивированного синтеза производят множество IDBC, содержащих олигионуклеотиды с заранее определенной уникальной последовательностью для каждого идентифицируемого ядра (т.е. микросферы). Поток IDBC из этого чипа комбинируется с потоком BP от аккумулятора BP в создаваемых в чипе каплях (то есть микрореакторах), которые генерируются с парами IDBC-BP для штрихкодирования и секвенирования. Детерминированное штрихкодирование полинуклеотидов BP позволяет сопоставлять данные секвенирования для этих полинуклеотидов с данными цитометрии и анализа изображений BP, из которых происходили полинуклеотиды.
В этом предпочтительном варианте осуществления, как показано на Фиг.3, где каждым компонент помечен буквой, система состоит из следующих компонентов:
Компонент A: Проточный цитометр с опциональным сортировщиком;
Компонент B: аккумулятор BP с устройством визуализации BP и референсных микросфер для подтверждения и отслеживания взаимного расположения BP и референсных микросфер;
Компонент C: GIDBC с ещё одним анализатором изображения референсных микрочастиц для подтверждения и отслеживания взаимного расположения кодирующих микрочастиц IDBC и референсных микросфер;
Компонент D: Чип для генерации капель вода-в-масле/микрореактора, где происходит высвобождение и штрихкодирование полинуклеотидов BP; и
Компонент E: проточный датчик IDBC референсных частиц для исправления ошибок, если относительное взаимное расположение неверно.
Система позволяет произвести полную интеграцию всех компонентов на одном чипе (как показано на рисунках 4A-C), комбинацию отдельных чипов, соединенных микрофлюидными линиями, и комбинацию основанных на чипах капиллярных и макроскопических устройств (как показано на Фиг. 3 и 5).
Авторы изобретения изготовили различные PDMS чипы (Фиг. 6A, B, D), включая прототипы отдельных GDBB (Фиг. 6C, 6E и 7A-8D), интегрированные в едином чипе инструменты (Фиг. 4A-C) и промежуточные варианты (например, чип со всеми компонентами, кроме GDBB (т.е. включающий микрофлюидику для FACS, аккумулятор BP, генератор капель и детектор синхронизации), (как показано на Фиг. 5).
Для изготовления чипов использовался двухслойный процесс мягкой литографии с 5 мкм-слоем ловушек и 40 мкм-слоем каналов для диаметра ядра от 5 до 10 мкм. Авторы изобретения изготовили литографическую маску для каждого слоя (Фиг.6A-6B), мастер-модель SU-8 (Фиг.6D) и слепки PDMS. Авторы изобретения вырезали отливки из PDMS по размеру отдельного чипа и приклеивали их к подложке из активированного стекла или отливке PDMS для изготовления чипов (Фиг. 6Е). Получающиеся в результате чипы имеют стеклянную сторону для оптического доступа и сторону PDMS для подключения жидкостных линий и магнитного воздействия (Фиг. 1B).
Подробное описание каждого компонента системы и его альтернатив приведено ниже:
(А) Цитометр/сортировщик
Проточный цитометр используется для проточного анализа фенотипа и сортировки интересующих BP. Хотя в системе возможно использовать большинство цитометров и других анализаторов частиц в потоке, предпочтительна схема на основе капилляров (Фиг. 3A) или схема на чипе (Фиг. 4A-5).
BP могут перемещаться через микрофлюидный канал с помощью внешнего давления, EOF, лазерной пульсации или других средств, хорошо известных специалистам в данной области. Авторы изобретения предлагают EOF и лазерную пульсацию в качестве основного движущего средства, потому что они быстро реагируют и не требуют движущихся частей. BP могут быть детектированы в канале или в кювете с гидродинамической фокусировкой с помощью LIF/LS. Детекция в канале проще в реализации и использовании, в то время как обнаружение в кювете с гидродинамической фокусировкой может обеспечить лучшее отношение сигнал / шум и более высокую пропускную способность. Сортировка может быть активирована с помощью флуоресцентного сигнала, сигнала светорассеяния или любого другого сигнала от детектора и выполняется с использованием перенаправления потока посредством EOF, лазерной пульсации или других средств, хорошо известных специалистам в данной области. Управление EOF проще в реализации, в то время как управление лазерными импульсами меньше зависит от химического состава поверхности стенок канала и обеспечивает лучшую пропускную способность. Как показано на Фиг. 3, часть образца, которая не соответствует критериям сортировки, выводится из системы как отходы.
(B) Аккумулятор клеток
Аккумулятор клеток опционально используется для (i) своевременной разделительной сортировки BP и генерации капель, чтобы избежать взаимных помех между драйверами потоков различных компонент, и (ii) визуализации BP и референсных частиц для коррекции ошибок.
BP загружаются в длинный микрофлюидный канал (Фиг. 3) или в более сложные микрофлюидные системы (аналогично показанным на Фиг. 7А-С и 9) и могут подаваться в том же порядке к компоненту D системы в любое удобное время. Во время загрузки BP с референсными частицами могут изображаться на чипе для проверки последовательности и объема загрузки через анализ количества BP между референсными частицами. Глубокий анализ изображения может также предоставить дополнительную информацию о фенотипе BP (например, форму ВР), дополняющую информацию, полученную с помощью проточного детектора.
Хотя этот компонент рекомендуется для расширения возможностей системы, он не является абсолютно необходимым. Если BP не используются, то они могут быть напрямую направлены в компонент D после сортировки из блока A. И наоборот, этот компонент с продвинутым анализом изображения может использоваться не только в дополнение, но и в качестве замены для блока A. В этом случае, фенотипические данные собираются исключительно посредством анализа изображения, хотя это также уменьшит возможности системы.
(C) GIDBC
В этом предпочтительном варианте осуществления ядром IDBC являются частицы в последовательном потоке, где каждая конкретная частица идентифицируется серийным номером (то есть положением в потоке) для отслеживания ее положения относительно других микрочастиц носителя и референсных частиц (Фиг. 3, 4А-C, 6C, 7A-C и 8A-D). Как описано выше, кодирующие олигонуклеотиды выращивают на частицах посредством фотоактивации (Фиг. 1). В этом варианте осуществления необходима обратимая иммобилизация частиц в «ловушках», расположенных в заранее определенных микрозонах на чипе. Эти ловушки расположены в микрофлюидном канале, который занимает как можно большую часть фотообработываемой площади чипа. Из описанных выше возможностей в этом предпочтительном варианте осуществления используются магнитные (Фиг. 1A-B, 2C, 4A-C, 6C, 6E и 8A-9) или гидродинамические (Фиг. 7A-C) ловушки.
Геометрия магнитных ловушек зависит от размера микросфер и гидродинамических свойств чипа. Для микросфер диаметром 8 мкм авторы изобретения изготовили чипы с шириной канала от 20 до 80 мкм, чтобы найти оптимальный баланс между загрузкой, контролем последовательности и сопротивлением засорению; круглые, треугольные и трапециевидные формы ловушек размером 5-20 мкм для оптимального контроля загрузки и высвобождения; и расстояние между ловушками от 50 до 100 мкм для оценки возможных оптических перекрестных помех и для контроля последовательности элюирования с изменением расстояния между ловушками (Фиг. 6A-E). В качестве альтернативного варианта осуществления авторы изобретения также изготовили чипы с набором гидродинамических ловушек (Фиг. 4A и Фиг. 7A-C). Чипы с диэлектрофорезными ловушками также рассматривались, но им был присвоен более низкий приоритет.
Оптимизация организации ловушек ядра в GIDBC. Для магнитной иммобилизации ловушки могут быть не только изотропными (то есть симметричными относительно потока среды), но также анизотропными для облегчения иммобилизации и высвобождения, в зависимости от направления потока среды, и упрощения управления вращением IDBC, если это необходимо. Для изотропных ловушек предпочтительна круговая геометрия, а для анизотропных ловушек предлагаются треугольные или трапециевидные ловушки (Фиг. 8A-D). Для улучшения покрытия микросфер олигонуклеотидными метками может быть реализовано вращение микросфер в ловушках. Вращение может быть вызвано посредством суперпозиции сдвигающей силы потока среды и силы захвата (магнитной, диэлектрофорезной и т. д.). Альтернативно, оптическое снятие защиты якорей может асимметрично экспонировать заряженные группы, создавая основанный на EOF импульс вращения. Кроме того, вращение электромагнитного поля может вызвать импульс, в случае соответствующей поляризуемости микрочастиц.
Оптимизация высвобождения микросфер в GIDBC. Высвобождение микросфер будет вызвано снятием силы захвата, при наличии сдвигающей силы потока среды. Однако при необходимости могут быть реализованы дополнительные средства контроля высвобождения и подавления нежелательной сорбции. К ним относятся (1) инвертированное или переменное поле, (2) реверсивный поток среды для анизотропных магнитных ловушек, как описано, и гидродинамические ловушки, (3) ультразвуковой драйвер и/или импульсное лазерное освещение, (4) обращение поверхностного заряда, (5) использование хаотропных агентов и (6) вращение микросфер.
Контроль последовательности микросфер в исходящем потоке в GIDBC: Для последовательного потока микросфер важно обеспечить сохранение взаимного порядка микросфер в исходящем потоке. Для мониторинга этого порядка авторы изобретения предлагают использовать оптически меченые микросферы в качестве референсных, для маркировки положений микросфер на чипе. С помощью этих референсных микросфер можно определить, остается ли количество немаркированных микросфер между оптически меченными в исходящем потоке одинаковым, и сравнить 2D-визуализацию оптически меченых референсных микросфер в ловушках в общей области фотообработки микросфер GIDBC с данными из LIF / LS проточного детектора (Фиг. 3-5).
Обработка микросфер с высокой производительностью. Для тысяч микросфер достаточно простейших одноканальных чипов (Фиг. 3, 4B, 4C, 6C, 6E, 8A-D) с элюцией под действием давления, лазерных импульсов или EOF. Тем не менее, для миллионов микросфер и более крупных чипов сопротивление потоку может быть слишком высоким для движения по одному каналу. В этом случае каналы могут заполняться параллельно и впоследствии элюироваться (см. пример с гидродинамическими ловушками на Фиг. 4А и Фиг. 7А-С). Более универсальная схема представлена на Фиг. 9.
В этой конструкции каналы могут заполняться и обрабатываться параллельно и элюироваться последовательно. Межканальные соединения потока могут быть заблокированы затвердевающей средой (например, фоторезистом) с последующим элюированием под давлением. Чипы, управляемые EOF, также допускают параллельную загрузку и обработку. Избыточные межканальные электрические соединения могут быть заблокированы изолирующей средой, что позволяет провести точно управляемое EOF элюирование независимо в каждом отдельном канале. Импульсная лазерная прокачка тоже может быть использована. Увеличение масштаба также потребовало бы более высокого разрешения оптической обработки, где могут возникнуть оптические перекрестные помехи. Подавление нежелательного распространения света может потребоваться посредством контроля показателя преломления, обработки поверхности для гашения света и увеличения размера области обработки.
(D) Генератор кодирующих микрореакторов в каплях.
Схемы чипа для генерации капель/микрореакторов вода-в-масле (WIO) для штрихкодирования полинуклеотидов BP хорошо разработаны в различных коммерческих приборах для mSCS. Вкратце, поток водной среды, содержащий кодирующие микросферы, анализируемые BP и все необходимые реагенты, встречает два потока масла (гидрофобной среды), подаваемых со стороны в месте соединения, с последующим локальным сужением канала (Фиг. 4А-С и Фиг. 5D), где образуются капли воды в масле.
Каждая капля должна содержать не более одной BP, потому что все полинуклеотиды в капле будут иметь одинаковый штрих-специфичный штрихкод, и полинуклеотиды из нескольких BP в одной капле не будут различаться после секвенирования. Капля без BP не вызывает никаких проблем, кроме потери эффективности. В результате, в обычно используемых чипах соотношение капель к BP поддерживается на высоком уровне, чтобы статистически уменьшить количество дублетов и мультиплетов BP на каплю. И, напротив, в предпочтительном варианте с использованием сортировщика/аккумулятора можно подавать BP в генератор капель более контролируемым образом, значительно уменьшая количество потерянных капель и IDBC. Этот уровень управления потоком достижим, потому что возбуждение среды лазерными импульсами или EOF имеет очень низкое время отклика на сигналы от проточного детектора LIF/LS, определяемое шириной импульсов или емкостью электродов, соответственно.
Каждая капля должна также содержать один IDBC. В капле без такой микросферы штрихкодирование невозможно, и информация о секвенировании BP будет потеряна. Если капля содержит несколько IDBC, то к полинуклеотидам из одного и той же BP будут применены разные штрихкоды, что вызовет проблемы с обработкой данных секвенирования при использовании традиционных чипов. Напротив, в предпочтительном варианте осуществления синхронизация и исправление ошибок (см. ниже) могут идентифицировать микрочастицы, участвующие в таких событиях, и обработка данных секвенирования не пострадает.
Каждая капля также должна содержать реагенты для лизиса ВР, лигирования полинуклеотида с кодирующим олигонуклеотидом, амплификации и иногда - обратной транскрипции. Эти реагенты могут подаваться потоком (-ами) водной среды, внутри IDBC или мигрировать в капли из масляной (гидрофобной) среды.
Как и в существующем mSCS, после совместного инкапсулирования BP, IDBC и реагентов в капле, BP лизируется для высвобождения полинуклеотидов, РНК подвергается обратной транскрипции (если необходимо), и лигазы используются для добавления кодирующего олигонуклеотида из IDBC к полинуклеотиду ВР. В связи с этим полинуклеотиды BP теперь несут штрихкод. Амплификация, если необходимо, может проводиться в капле или после слияния капель и удаления масляной (гидрофобной) среды при подготовке к секвенированию.
(E) Вспомогательный проточный детектор для синхронизации и исправления ошибок.
Упрощенный вторичный проточный детектор LIF/LS предназначен для анализа капель в исходящем потоке (Фиг. 3, Фиг. 4A-C, Фиг. 5), а именно для обнаружения и подсчета оптически меченых референсных микросфер. При использовании тех же принципов что и в LIF/LS-детекторе цитометра/сортировщика, этому детектору требуется один канал детекции светорассеяния и два канала обнаружения флуоресцентной (или SERS) метки, для обнаружения референсных микросфер, поэтому чувствительность может быть низкой. Скорость сбора данных должна соответствовать скорости генерации капель. Простейший анализ изображения также может быть полезным для этого детектора. В результате для этого компонента достаточно малобюджетных решений, таких как трехцветная веб-камера. Использование данных, полученных в этом блоке, описано ниже в разделе синхронизации и исправления ошибок.
Синхронизация и исправление ошибок с использованием оптических и генных меток. Связывание базы данных кодирующих последовательностей из GIDBC с цитометрическими данными подвержено ошибкам и создает риск для анализа. Ошибки могут возникать из-за обработки микросфер (то есть GIDBC) в чипе, потому что микрозоны синтеза олигонуклеотидов могут не содержать ни одной микросферы, содержать несколько микросфер, или микросферы могут сорбироваться вне микрозоны синтеза. Ошибки также могут быть вызваны чипом сопряжения IDBC и ВР в капле, потому что соотношение одной BP к одному IDBC на каплю может быть скомпрометировано. Ошибки могут возникать в проточном цитометре, поскольку дублеты или мультиплеты частиц могут быть обнаружены как одно событие. В каждом из этих случаев эти ошибки будут проявляться в несоответствии индексов в базе данных кодирующих последовательностей и данных цитометрии (то есть данных фенотипа, связанных с каждой BP и отслеживаемых по последовательности BP внутри системы). Эти несоответствия должны быть обнаружены и исправлены. Авторы изобретения предлагают два независимых механизма коррекции: перед секвенированием и после секвенирования.
Для способа после секвенирования, частицы (например, референсные частицы, липосомы), содержащие известные полинуклеотидные метки и оптические метки, могут быть добавлены к анализируемым ВР. Любое несоответствие между ожидаемой последовательностью референсных частиц в потоке ВР (на основе данных FC и изображения аккумулятора BP) и последовательностью помеченных частиц (IDBC) может быть обнаружено после секвенирования. Затем можно применить соответствующую коррекцию.
Для способа пред секвенирования оптически помеченные референсные частицы могут быть добавлены в IDBC (один цвет) и в BP (другой цвет), и проанализированы с помощью вторичного датчика потока (Фиг. 3-5), контролирующего несоответствия. Для IDBC он будет измерять расстояние (количество немеченных IDBC) между отмеченными (референсными), и сравнивать это расстояние с исходным референсным эталонным изображением GIDBC. Для BP он будет измерять, соответствует ли количество обнаруженных и отсортированных для кодирования кодирующих BP (на основе проточного детектора и анализа изображения аккумулятора ВР) количеству ВР между референсными частицами. Применяемые параллельно, эти методы обеспечивают надежное исправление ошибок.
Альтернативные варианты осуществления:
В альтернативных вариантах осуществления используются другие идентифицируемые ядра. Например, когда массив частиц или микрозон на чипе используется в качестве идентифицируемого ядра, где идентичность конкретного микрозоны определяется координатами микрозоны, это изобретение может использоваться с чипами, способными ограничивать и локализовывать реакцию биологических частиц ВР с микрозонами поверхности, содержащими кодирующие олигонуклеотиды. Такие микрозоны включают, но не ограничиваются, микролунками на поверхности чипа, которые ограничивают отдельные ВР, или гелевыми барьерами, вводимыми оптически, электромагнитно или механически. В этом подходе анализ изображений используется для доступа к фенотипам ВР вместо проточных методов.
Когда IDBC со встроенными уникальными идентификационными метками, которые могут быть идентифицированы в потоке (например, RFID или оптические метки, как описано выше), используются в качестве идентифицируемых ядер, олигонуклеотиды могут быть выращены с использованием всех упомянутых методов, но синтез с отслеживаемым смешиванием является наиболее подходящим (как описано выше, см. Фиг. 2D). Раствор (Суспензия) этих IDBC вместе с соответствующим проточным детектором идентификатора может быть использована в предпочтительном варианте конструкции прибора вместо GIDBC на основе чипа, что устраняет необходимость многоэтапного выращивания олигонуклеотида для каждой загрузки частиц GIDBC.
Настоящее изобретение было описано со ссылкой на некоторые предпочтительные и альтернативные варианты осуществления, которые предназначены только для примера и не ограничивают полный объем настоящего изобретения. Хотя элементы предлагаемого устройства являются синергетическими, некоторые из них могут быть устранены без полной потери функциональности. Например, сортировка клеток на основе проточной цитометрии может быть устранена за счет более высокого потребления IDBC и загрузки секвенсора. Проточная цитометрия может быть устранена и заменена анализом изображения аккумулятора биологических частиц (как описано выше). Даже проточный цитометр, генератор капель и основанные на частицах IDBC можно исключить, если совместить GIDBC и аккумулятор биологических частиц, а массив IDBC напечатать непосредственно на поверхности чипа.
В предпочтительных или альтернативных вариантах осуществления нет необходимости выращивать кодирующие олигонуклеотиды на идентифицируемых ядрах перед каждым использованием системы. Вместо этого IDBC могут быть приготовлены заранее или даже поставлены в виде одноразового картриджа для анализа при использовании (i) заранее загруженного IDBC одноразового чипа-GIDBC; (ii) чипа с массивом микрозон поверхности, содержащих кодирующие олигонуклеотиды; или (iii) суспензии IDBC с идентифицируемыми в потоке ядрами, с соответствующей базой данных, связывающей идентификатор ядра (серийный номер, положение или RFID/оптические метки) каждого элемента IDBC с последовательностью его кодирующих олигонуклеотидов, как описано выше.
Источники
1. Kimmerling, R.J., et al., A microfluidic platform enabling single-cell RNA-seq of multigenerational lineages. Nat Commun, 2016. 7: p. 10220.
2. Andreyev, D., Development of models and analytical approaches for individual mitochondria, in Chemistry. 2007 University of Minnesota: Minneapolis, MN. p. 208.
3. Andreyev, D.S. and E.A. Arriaga, Fabrication of perforated sub-micron silica shells. Scr Mater, 2007. 57(10): p. 957-959.
4. Lan, F., et al., Single-cell genome sequencing at ultra-high-throughput with microfluidic droplet barcoding. Nat Biotechnol, 2017. 35(7): p. 640-646.
5. Zheng, G.X., et al., Massively parallel digital transcriptional profiling of single cells. Nat Commun, 2017. 8: p. 14049.
6. Jaitin, D.A., et al., Massively parallel single-cell RNA-seq for marker-free decomposition of tissues into cell types. Science, 2014. 343(6172): p. 776-9.
7. Stoeckius, M., et al., Simultaneous epitope and transcriptome measurement in single cells. Nat Methods, 2017. 14(9): p. 865-868.
8. Bongso, A., C.Y. Fong, and K. Gauthaman, Taking stem cells to the clinic: Major challenges. J Cell Biochem, 2008. 105(6): p. 1352-60.
9. Saini, N., et al., The Impact of Environmental and Endogenous Damage on Somatic Mutation Load in Human Skin Fibroblasts. PLoS Genet, 2016. 12(10): p. e1006385.
10. Herberts, C.A., M.S. Kwa, and H.P. Hermsen, Risk factors in the development of stem cell therapy. J Transl Med, 2011. 9: p. 29.
11. Serakinci, N., U. Fahrioglu, and R. Christensen, Mesenchymal stem cells, cancer challenges and new directions. Eur J Cancer, 2014. 50(8): p. 1522-30.
12. Hong, I.S., H.Y. Lee, and K.S. Kang, Mesenchymal stem cells and cancer: friends or enemies? Mutat Res, 2014. 768: p. 98-106.
13. Glinsky, G.V., Activation of endogenous human stem cell-associated retroviruses (SCARs) and therapy-resistant phenotypes of malignant tumors. Cancer Lett, 2016. 376(2): p. 347-59.
14. Porta, C., E. Riboldi, and A. Sica, Mechanisms linking pathogens-associated inflammation and cancer. Cancer Lett, 2011. 305(2): p. 250-62.
15. Rom, J.S., et al., Impact of Staphylococcus aureus regulatory mutations that modulate biofilm formation in the USA300 strain LAC on virulence in a murine bacteremia model. Virulence, 2017. 8(8): p. 1776-1790.
16. Leites, E.P. and V.A. Morais, Mitochondrial quality control pathways: PINK1 acts as a gatekeeper. Biochem Biophys Res Commun, 2017.
17. Grosso, R., C.M. Fader, and M.I. Colombo, Autophagy: A necessary event during erythropoiesis. Blood Rev, 2017. 31(5): p. 300-305.
18. Neill, T., L. Schaefer, and R.V. Iozzo, Oncosuppressive functions of decorin. Mol Cell Oncol, 2015. 2(3): p. e975645.
19. Salgado, J., B. Honorato, and J. García-Foncillas, Review: mitochondrial defects in breast cancer. Clin Med Oncol, 2008. 2: p. 199-207.
20. Stefano, G.B. and R.M. Kream, Mitochondrial DNA heteroplasmy in human health and disease. Biomed Rep, 2016. 4(3): p. 259-262.
21. Chatterjee, A., E. Mambo, and D. Sidransky, Mitochondrial DNA mutations in human cancer. Oncogene, 2006. 25(34): p. 4663-74.
22. He, Y., et al., Heteroplasmic mitochondrial DNA mutations in normal and tumour cells. Nature, 2010. 464(7288): p. 610-4.
23. Parr, R.L., et al., Mitochondrial DNA as a potential tool for early cancer detection. Hum Genomics, 2006. 2(4): p. 252-7.
24. Sukhanova, A. and I. Nabiev, Fluorescent nanocrystal-encoded microbeads for multiplexed cancer imaging and diagnosis. Crit Rev Oncol Hematol, 2008. 68(1): p. 39-59.
25. Hintersteiner, M., et al., Single bead labeling method for combining confocal fluorescence on-bead screening and solution validation of tagged one-bead one-compound libraries. Chem Biol, 2009. 16(7): p. 724-35.
26. Sha, M.Y., et al., Surface-enhanced Raman scattering tags for rapid and homogeneous detection of circulating tumor cells in the presence of human whole blood. J Am Chem Soc, 2008. 130(51): p. 17214-5.
27. Wood, D.K., et al., A feasible approach to all-electronic digital labeling and readout for cell identification. Lab Chip, 2007. 7(4): p. 469-74.
28. Nuwaysir, E.F., et al., Gene expression analysis using oligonucleotide arrays produced by maskless photolithography. Genome Res, 2002. 12(11): p. 1749-55.
29. Zybailov, B., et al., Analysis of Protein-protein Interaction Interface between Yeast Mitochondrial Proteins Rim1 and Pif1 Using Chemical Cross-linking Mass Spectrometry. J Proteomics Bioinform, 2015. 8(11): p. 243-252.
30. Zybailov, B., Q. Sun, and K. van Wijk, Workflow for large scale detection and validation of peptide modifications by RPLC-LTQ-Orbitrap: application to the Arabidopsis thaliana leaf proteome and an online modified peptide library. Anal Chem, 2009. 81(19): p. 8015-24.
31. Zybailov, B., et al., Correlation of relative abundance ratios derived from peptide ion chromatograms and spectrum counting for quantitative proteomic analysis using stable isotope labeling. Anal Chem, 2005. 77(19): p. 6218-24.
32. Zybailov, B., et al., Recruitment of a foreign quinone into the A(1) site of photosystem I. II. Structural and functional characterization of phylloquinone biosynthetic pathway mutants by electron paramagnetic resonance and electron-nuclear double resonance spectroscopy. J Biol Chem, 2000. 275(12): p. 8531-9.
33. Shinkarev, V., et al., Modeling of the P700+ charge recombination kinetics with phylloquinone and plastoquinone-9 in the A1 site of photosystem I. Biophys J, 2002. 83(6): p. 2885-97.
34. Semenov, A., et al., Recruitment of a foreign quinone into the A1 site of photosystem I. Altered kinetics of electron transfer in phylloquinone biosynthetic pathway mutants studied by time-resolved optical, EPR, and electrometric techniques. J Biol Chem, 2000. 275(31): p. 23429-38.
35. Johnson, R.D., et al., Analysis of mitochondria isolated from single cells. Anal Bioanal Chem, 2007. 387(1): p. 107-18.
36. Andreyev, D. and E.A. Arriaga, Simultaneous laser-induced fluorescence and scattering detection of individual particles separated by capillary electrophoresis. Anal Chem, 2007. 79(14): p. 5474-8.
37. Ahmadzadeh, H., et al., Capillary electrophoresis reveals changes in individual mitochondrial particles associated with skeletal muscle fiber type and age. J Gerontol A Biol Sci Med Sci, 2006. 61(12): p. 1211-8.
38. Shalgunov, V., et al., Comprehensive study of the drug delivery properties of poly(l-lactide)-poly(ethylene glycol) nanoparticles in rats and tumor-bearing mice. J Control Release, 2017. 261: p. 31-42.
39. Shields, C.W., C.D. Reyes, and G.P. López, Microfluidic cell sorting: a review of the advances in the separation of cells from debulking to rare cell isolation. Lab Chip, 2015. 15(5): p. 1230-49.
40. Chen, Y., et al., 3D pulsed laser-triggered high-speed microfluidic fluorescence-activated cell sorter. Analyst, 2013. 138(24): p. 7308-15.
41. Dijkink, R. and C.D. Ohl, Laser-induced cavitation based micropump. Lab Chip, 2008. 8(10): p. 1676-81.
42. Wu, T.H., et al., Pulsed laser triggered high speed microfluidic fluorescence activated cell sorter. Lab Chip, 2012. 12(7): p. 1378-83.
43. Gijs, M.A.M., Magnetic bead handling on-chip: new opportunities for analytical applications. Microfluid Nanofluid, 2004. 1: p. 22-40.
44. Rosenthal, A. and J. Voldman, Dielectrophoretic traps for single-particle patterning. Biophys J, 2005. 88(3): p. 2193-205.
45. Qiao, W., G. Cho, and Y.H. Lo, Wirelessly powered microfluidic dielectrophoresis devices using printable RF circuits. Lab Chip, 2011. 11(6): p. 1074-80.
46. Noda, H., et al., Automated bead alignment apparatus using a single bead capturing technique for fabrication of a miniaturized bead-based DNA probe array. Anal Chem, 2003. 75(13): p. 3250-5.
47. Yeo, J.C., Z. Wang, and C.T. Lim, Microfluidic size separation of cells and particles using a swinging bucket centrifuge. Biomicrofluidics, 2015. 9(5): p. 054114.
48. Friend, J. and L. Yeo, Fabrication of microfluidic devices using polydimethylsiloxane. Biomicrofluidics, 2010. 4(2).
49. Eddings, M.A., M.A. Johnson, and B.K. Gale, Determining the optimal PDMS–PDMS bonding technique for microfluidic devices. Journal of Micromechanics and Microengineering, 2008. 18(6): p. 067001.
50. Estevez, M.-C., M. Alvarez, and L.M. Lechuga Integrated optical devices for lab-on-a-chip biosensing applications. Laser Photonics Rev, 2012. 6(4): p. 463-487.
51. Sinn, I., et al., Asynchronous magnetic bead rotation (AMBR) biosensor in microfluidic droplets for rapid bacterial growth and susceptibility measurements. Lab Chip, 2011. 11(15): p. 2604-11.
52. van Pelt, S., et al., Flow-orthogonal bead oscillation in a microfluidic chip with a magnetic anisotropic flux-guide array. Biomed Microdevices, 2011. 13(2): p. 353-9.
53. Iranmanesh, I., et al., Acoustic micro-vortexing of fluids, particles and cells in disposable microfluidic chips. Biomed Microdevices, 2016. 18(4): p. 71.
54. Zwaan, E., et al., Controlled cavitation in microfluidic systems. Phys Rev Lett, 2007. 98(25): p. 254501.
55. Deyholos, M.K. and D.W. Galbraith, High-density microarrays for gene expression analysis. Cytometry, 2001. 43(4).
56. Ong, S.G., et al., Evaluation of Extrusion Technique for Nanosizing Liposomes. Pharmaceutics, 2016. 8(4).
57. Wolken, G.G. and E.A. Arriaga, Simultaneous measurement of individual mitochondrial membrane potential and electrophoretic mobility by capillary electrophoresis. Anal Chem, 2014. 86(9): p. 4217-26.
58. Cottet-Rousselle, C., et al., Cytometric assessment of mitochondria using fluorescent probes. Cytometry A, 2011. 79(6): p. 405-25.
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБ ИЗГОТОВЛЕНИЯ НАБОРОВ НУКЛЕОТИДНЫХ И/ИЛИ ИНЫХ МОЛЕКУЛЯРНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ С ИСПОЛЬЗОВАНИЕМ МИКРОЧАСТИЦ, НЕСУЩИХ МЕТКИ, ВЗАИМНО-ОДНОЗНАЧНО СВЯЗАННЫЕ С ИЗГОТАВЛИВАЕМЫМИ ПОСЛЕДОВАТЕЛЬНОСТЯМИ | 2022 |
|
RU2784192C1 |
| СПОСОБ МОДИФИКАЦИИ БОЛЬШИХ НАБОРОВ МИКРОЧАСТИЦ, НЕСУЩИХ УНИКАЛЬНЫЕ ФЛУОРЕСЦЕНТНЫЕ МЕТКИ | 2022 |
|
RU2829216C2 |
| СПОСОБ ПОЛУЧЕНИЯ ГИДРОГЕЛЯ С ТЕРМООТЩЕПЛЯЕМЫМИ НУКЛЕИНОВЫМИ КИСЛОТАМИ | 2022 |
|
RU2826936C2 |
| СЕКВЕНИРОВАНИЕ ПОЛИНУКЛЕОТИДНЫХ БИБЛИОТЕК С ВЫСОКОЙ ПРОПУСКНОЙ СПОСОБНОСТЬЮ И АНАЛИЗ ТРАНСКРИПТОМОВ | 2018 |
|
RU2790291C2 |
| КОНЪЮГАТЫ АФФИННАЯ ЧАСТЬ-ОЛИГОНУКЛЕОТИД И ИХ ПРИМЕНЕНИЯ | 2016 |
|
RU2778754C2 |
| ВЫСОКОПРОИЗВОДИТЕЛЬНОЕ СЕКВЕНИРОВАНИЕ ОДИНОЧНОЙ КЛЕТКИ СО СНИЖЕННОЙ ОШИБКОЙ АМПЛИФИКАЦИИ | 2019 |
|
RU2744175C1 |
| ГОМОЛОГИ (ATR) ПОЛИПЕПТИДА rad3, ПОЛИНУКЛЕОТИДЫ И СВЯЗАННЫЕ С НИМИ СПОСОБЫ И МАТЕРИАЛЫ | 1996 |
|
RU2252256C2 |
| СПОСОБ И КОМПОЗИЦИЯ ДЛЯ АНАЛИЗА КЛЕТОЧНЫХ КОМПОНЕНТОВ | 2016 |
|
RU2717491C2 |
| БЕЛОК IFNAB-BPII, ОБЛАДАЮЩИЙ СПОСОБНОСТЬЮ СВЯЗЫВАТЬ ИНТЕРФЕРОН-α/β, ЕГО ПРЕДШЕСТВЕННИК И СЛИТЫЕ БЕЛКИ, СПОСОБ ПОЛУЧЕНИЯ IFNAB-BPII, МОЛЕКУЛЫ ДНК, ЭКСПРЕССИРУЮЩИЙ ВЕКТОР, ФАРМАЦЕВТИЧЕСКАЯ КОМПОЗИЦИЯ | 1995 |
|
RU2232811C2 |
| ПОЛИНУКЛЕОТИДЫ И ПОЛИПЕПТИДЫ ФАГА φ-mru, И ИХ ПРИМЕНЕНИЕ | 2008 |
|
RU2520738C2 |

Группа изобретений относится к области биотехнологии. Предложены система и способ анализа фенотипа и последовательности полинуклеотидов биологической частицы. Система содержит анализатор фенотипа биологической частицы, генератор идентифицируемого олигонуклеотидного кодирующего носителя, микрореактор, секвенатор предварительно определенной последовательности кодирующего олигонукеголеотида и последовательности полинуклеотидов биологической частицы. Способ включает оптический анализ биологической частицы для определения фенотипа, запись фенотипа, генерирование носителя кодирующих олигонуклеотидов, помещение биологической частицы и носителя в микрореактор, лизис биологической частицы, лигирование кодирующих олигонуклеотидов с полинуклеотидами биологической частицы, секвенирование для определения последовательности полинуклеотидов биологической частицы и сопоставление фенотипа биологической частицы с последовательностью полинуклеотидов биологической частицы. Изобретения обеспечивают точное определение генома/транскриптома и фенотипа для каждой биологической частицы. 2 н. и 13 з.п. ф-лы, 24 ил.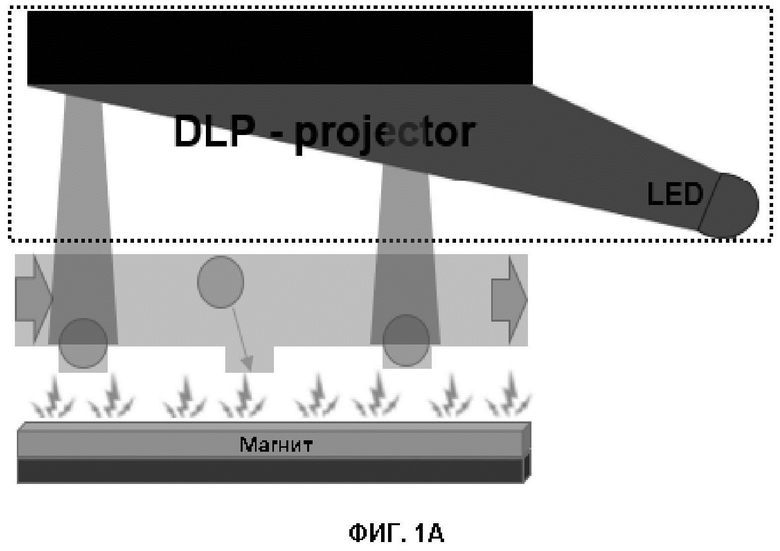
1. Система для анализа фенотипа и последовательности полинуклеотидов биологической частицы, которая содержит:
- анализатор фенотипа, где указанный анализатор фенотипа предназначен для определения фенотипа указанной биологической частицы;
- генератор идентифицируемого олигонуклеотидного кодирующего носителя, где кодирующий олигонуклеотид с заранее известной последовательностью прикреплен к указанному носителю, используется индивидуальный носитель для каждой биологической частицы и применяется уникальный и предварительно определенный штрихкод для каждого полинуклеотида каждой биологической частицы;
- микрореактор, выполненный с возможностью приема указанной биологической частицы из указанного анализатора фенотипа и приема указанного носителя кодирующих олигонуклеотидов из указанного генератора;
- секвенатор указанной предварительно определенной последовательности указанного кодирующего олигонукеголеотида и указанной последовательности полинуклеотидов указанной биологической частицы после того, как указанный кодирующий олигонуклеотид лигируют с указанными полинуклеотидами указанной биологической частицы.
2. Система по п. 1, отличающаяся тем, что анализатор фенотипа представляет собой проточный цитометр.
3. Система по п. 1, отличающаяся тем, что анализатор фенотипа представляет собой микроскоп.
4. Система по п. 1, отличающаяся тем, что носитель представляет собой магнитную частицу.
5. Система по п. 1, отличающаяся тем, что носитель представляет собой микроячейку на поверхности чипа.
6. Система по п. 1, отличающаяся тем, что содержит аккумулятор биологических частиц между указанным анализатором фенотипа и указанным микрореактором.
7. Способ анализа фенотипа и последовательности полинуклеотидов биологической частицы, включающий стадии:
- оптического анализа указанной биологической частицы для определения фенотипа указанной биологической частицы;
- записи указанного фенотипа указанной биологической частицы;
- генерирования носителя кодирующих олигонуклеотидов, где кодирующий олигонуклеотид предварительно определенной последовательности прикреплен к указанному носителю, используется индивидуальный носитель для каждой биологической частицы и применяется уникальный и предварительно определенный штрихкод для каждого полинуклеотида каждой биологической частицы;
- помещения указанной биологической частицы и указанного носителя кодирующих олигонуклеотидов в микрореактор;
- лизиса указанной биологической частицы для высвобождения указанных полинуклеотидов указанной биологической частицы;
- лигирования указанных кодирующих олигонуклеотидов с указанными полинуклеотидами указанной биологической частицы для образования генетического комплекса;
- секвенирования указанного генетического комплекса для определения указанной последовательности указанных полинуклеотидов указанной биологической частицы;
- сопоставления указанного фенотипа указанной биологической частицы с указанной последовательностью указанных полинуклеотидов указанной биологической частицы.
8. Способ по п. 7, отличающийся тем, что указанная биологическая частица и указанный носитель кодирующих олигонуклеотидов инкапсулированы в капле в указанном микрореакторе.
9. Способ по п. 7, отличающийся тем, что проточный цитометр выполняет этап оптического анализа указанной биологической частицы для определения фенотипа указанной биологической частицы.
10. Способ по п. 7, отличающийся тем, что указанная биологическая частица подается в аккумулятор биологических частиц до того, как указанная биологическая частица помещается в указанный микрореактор.
11. Способ по п. 7, отличающийся тем, что содержит этап анализа исходящего из указанного микрореактора на наличие ошибок.
12. Способ по п. 7, отличающийся тем, что указанная стадия генерирования носителя кодирующих олигонуклеотидов включает выращивание олигонуклеотида на частице.
13. Способ по п. 12, отличающийся тем, что стадия выращивания олигонуклеотида на частице включает фотоактивацию.
14. Способ по п. 12, отличающийся тем, что включает стадию иммобилизации указанной частицы в ловушке на чипе.
15. Способ по п. 7, отличающийся тем, что стадия генерирования носителя кодирующих олигонуклеотидов включает выращивание олигонуклеотида на чипе.
| Многоступенчатая активно-реактивная турбина | 1924 |
|
SU2013A1 |
| Приспособление для суммирования отрезков прямых линий | 1923 |
|
SU2010A1 |
| Токарный резец | 1924 |
|
SU2016A1 |
| Токарный резец | 1924 |
|
SU2016A1 |
| СПОСОБ ОПРЕДЕЛЕНИЯ ПОСЛЕДОВАТЕЛЬНОСТИ НУКЛЕИНОВОЙ КИСЛОТЫ (ВАРИАНТЫ) И НАБОР ДЛЯ ИСПОЛЬЗОВАНИЯ ПРИ ОПРЕДЕЛЕНИИ ПОСЛЕДОВАТЕЛЬНОСТИ НУКЛЕИНОВОЙ КИСЛОТЫ | 1994 |
|
RU2143004C1 |
Авторы
Даты
2021-09-29—Публикация
2018-09-21—Подача