Область техники, к которой относится изобретение
Настоящее изобретение относится к анти-PD-L1 антителу для детекции PD-L1.
Уровень техники
Злокачественная меланома, возникающая из меланоцитов, является одной из наиболее часто встречающихся злокачественных опухолей в ротовой полости собак (непатентный документ № 1: Todoroff et al., J Am Vet Med Assoc. 1979 Sep 15; 175(6):567-71). Поскольку этот тип меланомы, как правило, имеет тенденцию к высокой инвазии и метастазированию, желательно иметь возможность ранней диагностики и лечения. С другой стороны, злокачественная меланома имеет широкое тканевое разнообразие и различную морфологию, например, эпителиальная, круглоклеточная меланома или меланома с морфологией фибросаркомы. Таким образом, злокачественная меланома относится к опухолям с трудной тканевой диагностикой. Хотя подтверждение наличия пигмента меланина важно для диагностики злокачественных меланом, большое их количество не имеет пигмента меланина и, иногда, диагноз не может быть поставлен только на основе гистологической оценки. Это привело к поиску диагностических маркеров, которые можно использовать в иммуногистохимии. Было описано, что среди таких маркеров, Melan A/MART-1, виментин, S100, нейрон-специфическая енолаза и тому подобное, могут быть эффективными (непатентный документ № 2: Ramos-Vara et al., Vet Pathol. 2000 Nov; 37(6):597-608). Однако даже Melan A/MART-1, наиболее широко используемый диагностический маркер, имеет положительный прогностический показатель не выше 60%, который отличается по разным данным (непатентный документ № 3: Koenig et al., Vet Pathol. 2001 Jul; 38(4):427-35). Из-за проблемы чувствительности эффективность Melan A/MART-1 для диагностики в практике остается спорной. Кроме того, Melan A/MART-1 не окрашивается в беспигментных меланомах (непатентный документ № 3: Koenig et al., Vet Pathol. 2001 Jul; 38(4):427-35), поэтому его применение в диагностике ограничена. В этих условиях желательно разработать высокочувствительные, новые маркеры для диагностики злокачественной меланомы.
Документы уровня техники
Непатентные документы
Непатентный документ № 1: Todoroff et al., J Am Vet Med Assoc. 1979 Sep 15; 175(6):567-71
Непатентный документ № 2: Ramos-Vara et al., Vet Pathol. 2000 Nov; 37(6):597-608).
Непатентный документ № 3: Koenig et al., Vet Pathol. 2001 Jul; 38(4):427-35
Раскрытие сущности изобретения
Задача, решаемая изобретением
Целью настоящего изобретения является получение антитела против PD-L1, способного окрашивать опухолевые клетки, такие как клетки меланомы.
Средства решения задачи
Авторы настоящего изобретения обнаружили ряд моноклональных антител, которые взаимодействуют с белком PD-L1 различных животных. Было обнаружено, что среди этих моноклональных антител крысиное анти-бычье PD-L1 моноклональное антитело (6С11-3А11) способно высокоэффекивно окрашивать опухолевые клетки меланомы. В настоящее время это моноклональное антитело используется для селекции собак-кандидатов для терапии химерными антителами. Исследуемое антитело против PD-L1 (6C11-3A11) также способно иммуногистохимически окрашивать белки PD-L1 овец, свиней и крупного рогатого скота. Кроме того, авторы настоящего изобретения определили CDR (определяющие комплементарность область) вариабельных областей антитела против PD-L1 (6C11-3A11). Настоящее изобретение было осуществлено на основании этих обнаружений.
Краткое описание изобретения
(1) Анти-PD-L1 антитело, содержащее (a) легкую цепь, содержащую CDR1 с аминокислотной последовательностью KSISKY (SEQ ID NO:1), CDR2 с аминокислотной последовательностью SGS и CDR3 с аминокислотной последовательностью QQHNEYPLT (SEQ ID NO:2) и (b) тяжелую цепь, содержащую CDR1 с аминокислотной последовательностью GYTFTDYI (SEQ ID NO:3), CDR2 с аминокислотной последовательностью INPDSGGN (SEQ ID NO:4) и CDR3 с аминокислотной последовательностью ARGITMMVVISHWKFDF (SEQ ID NO:5).
(2) Антитело по (1) выше, которое получено у крысы.
(3) Антитело по (2) выше, которое представляет собой крысиное анти-бычье PD-L1 антитело.
(4) Антитело по (3) выше, где вариабельная область легкой цепи имеет аминокислотную последовательность SEQ ID NO:6, и вариабельная область тяжелой цепи имеет аминокислотную последовательность SEQ ID NO:7.
(5) Антитело по любому из (1)-(4) выше, где константная область легкой цепи имеет аминокислотную последовательность константной области легкой цепи каппа.
(6) Антитело по любому из (1)-(5) выше, где константная область тяжелой цепи имеет аминокислотную последовательность константной области IgG2a.
(7) Антитело по (5) или (6) выше, где константная область легкой цепи имеет аминокислотную последовательность SEQ ID NO:8, 10-12 и константная область тяжелой цепи имеет аминокислотную последовательность SEQ ID NO:9 или 13.
(8) Антитело по любому из (1)-(7) выше, которое имеет структуру четырех цепей, содержащую две легкие цепи и две тяжелые цепи.
(9) Композиция для детекции PD-L1, содержащая антитело по любому из (1)-(8) выше в качестве активного ингредиента.
(10) Композиция по (9) выше для применения в диагностике злокачественных новообразований и/или воспалительных процессов.
(11) Композиция по (10) выше, где злокачественные новообразования и /или воспалительные процессы выбраны из группы, включающей неопластические заболевания, лейкоз, болезнь Джона, анаплазмоз, бактериальный мастит, грибковый мастит, инфекции, вызванные микоплазмой (например, мастит, вызванный микоплазмой, пневмония, вызванная микоплазмой, или тому подобное), туберкулез, инфекцию, вызванную Theileria orientalis, криптоспоридоз, кокцидиоз, трипаносомоз и лейшманиоз.
(12) Композиция по (9) выше для применения в селекции животных, подходящих для терапии анти-PD-L1 антителами.
(13) ДНК, кодирующая анти-PD-L1 антитело по (1) выше.
(14) Вектор, содержащий ДНК по (13) выше.
(15) Клетка-хозяин, трансформированная вектором по (14) выше.
(16) Способ получения антитела, включающий культивирование клетки-хозяина по (15) выше и сбор анти-PD-L1 антитела выше из полученной культуры.
(17) ДНК, кодирующая легкую цепи анти-PD-L1 антитела, где указанная легкая цепь содержит CDR1 с аминокислотной последовательностью KSISKY (SEQ ID NO:1), CDR2 с аминокислотной последовательностью SGS, и CDR3 с аминокислотной последовательностью QQHNEYPLT (SEQ ID NO:2).
(18) ДНК, кодирующая тяжелую цепи анти-PD-L1 антитела, где указанная тяжелая цепь содержит CDR1 с аминокислотной последовательностью GYTFTDYI (SEQ ID NO:3), CDR2 с аминокислотной последовательностью INPDSGGN (SEQ ID NO:4), и CDR3 с аминокислотной последовательностью ARGITMMVVISHWKFDF (SEQ ID NO:5).
Эффект изобретения
Согласно настоящему изобретению, было получено новое анти-PD-L1 антитело, способное окрашивать опухолевые клетки, такие как клетки меланомы.
В настоящем описании приведено содержание описания и/или чертежей японской патентной заявки № 2017-61389, на основании которой заявляется приоритет настоящей патентной заявки.
Краткое описание чертежей
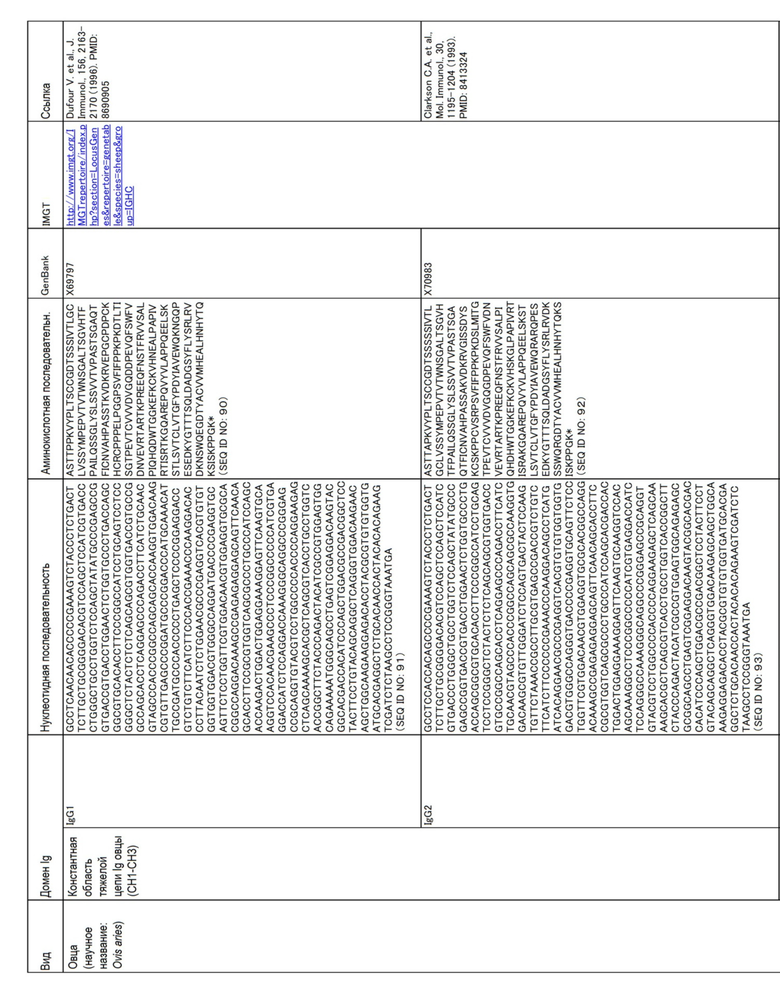
[ФИГ.1] Специфичность связывания крысиного анти-бычьего PD-L1 антитела 6С11-3А11. Крысиный анти-бычий PD-L1 антитела 6C11-3A11 не связывались с клетками, экспрессирующими EGFP, но специфически связывались с клетками, экспрессирующими PD-L1-EGFP, собаки.
[ФИГ.2] Спрогнозированные области CDR крысиного анти-бычьего PD-L1 антитела 6С11-3А11. Показаны области CDR1, CDR2 и CDR3 в вариабельной области легкой цепи и вариабельной области тяжелой цепи крысиного анти-бычьего PD-L1 антитела 6С11-3А11.
[ФИГ.3] Фотографии сравнительного иммуногистохимического окрашивания меланомы собак. Слева: окрашено коммерческим антителом (антитело MelanA). Опухолевые клетки окрашивались очень слабо. Справа: окрашено антителом против PD-L1, 6C11-3A11, полученным авторами настоящего изобретения. Опухолевые клетки были значительно окрашены.
[ФИГ.4] Фотография иммуногистохимического окрашивания меланомы собак.
[ФИГ.5-1] Фотографии иммуногистохимического окрашивания других опухолей. Слева вверху: лимфома собак. Справа вверху: остеосаркома собак. Слева внизу: 1-ый пример почечно-клеточного рака у собак. Справа внизу: 2-ой пример почечно-клеточного рака у собак.
[ФИГ.5-2] Фотографии иммуногистохимического окрашивания других опухолей. Слева: случай плоскоклеточного рака у собак. Справа: случай фибросаркомы собак.
[ФИГ.6] Фотографии иммуногистохимического окрашивания листериоза овец. Слева: Фотография с окрашиванием PD-L1 поражения головного мозга при листериозе с неврологическими симптомами у овец. Справа: увеличенная фотография левого изображения.
[ФИГ.7] Фотографии иммуногистохимического окрашивания инфекций у свиней. Слева: инфекция цирковирусом 2-ого типа у свиней. Справа: пневмония, вызванная микоплазмой, у свиней.
[ФИГ.8] Выравнивание аминокислотных последовательностей константной области каппа цепи (легкая цепь) Ig крысы.
[ФИГ.9] Выравнивание аминокислотных последовательностей константной области цепи IgG2a (тяжелая цепь) крысы.
[ФИГ.10] Схематическое изображение вектора pDC6 и химерного крысиного-человеческого анти-PD-L1 антитела.
[ФИГ.11] Связывание крысиных анти-бычьих PD-L1 антител 6C11-3A11 и 6G7-Е1 c клетками, экспрессирующим PD-L1-EGFP, собаки. 6С11-3A11 специфически связывается с клетками, экспрессирующими PD-L1-EGFP, собаки.
[ФИГ.12] Фотография иммуногистохимического окрашивания плоскоклеточного рака кожи, аденокарциномы носа и переходно-клеточного рака у собак. Не было обнаружено специфических сигналов с помощью 6G7-E1. Опухолевые клетки окрашивали 6C11-3A11.
[ФИГ.13] Фотографии иммуногистохимического окрашивания рака анального мешка, саркомы мягких тканей и остеосаркомы у собак. При раке анального мешка и саркоме мягких тканей, не было детектировано специфических сигналов с помощью 6G7-E1, но опухолевые клетки окрашивались 6C11-3A11. При остеосаркоме оба антитела окрашивали опухолевые клетки, но более сильные сигналы были получены с помощью 6C11-3A11.
[ФИГ.14] Фотографии иммуногистохимического окрашивания злокачественной меланомы полости рта, аденокарциномы молочной железы, гистиоцитарной саркомы, диффузной В-крупноклеточной лимфомы и трансмиссивной венерической опухоли у собак с использованием 6C11-3A11. В опухолях, отличных от трансмиссивной венерической опухоли, опухолевые клетки окрашивались PD-L1.
[ФИГ.15] Связывание крысиного анти-бычьего PD-L1 антитела 6C11-3A11 с бычьим клетками, экспрессирующими PD-L1-EGFP. 6С11-3A11 специфически связывается с бычьими клетками, экспрессирующими PD-L1-EGFP.
[ФИГ.16] Фотография иммуногистохимического окрашивания поражений подвздошной кишки крупного рогатого скота, вызванных естественным путем и экспериментально Mycobacterium avium Mycobacterium avium subsp. paratuberculosis, используя (a) 6C11-3A11 и (b) окрашивание по Цилю-Нильсену).
Варианты осуществления изобретения
Ниже настоящее изобретение описано подробно.
Настоящее изобретение относится к анти-PD-L1 антителу, содержащему (a) легкую цепь, содержащую CDR1 с аминокислотной последовательностью KSISKY (SEQ ID NO:1), CDR2 с аминокислотной последовательностью SGS, и CDR3 с аминокислотной последовательностью QQHNEYPLT (SEQ ID NO:2), и (b) тяжелую цепь, содержащую CDR1 с аминокислотной последовательностью GYTFTDYI (SEQ ID NO:3), CDR2 с аминокислотной последовательностью INPDSGGN (SEQ ID NO:4), и CDR3 с аминокислотной последовательностью ARGITMMVVISHWKFDF (SEQ ID NO:5).
CDR1, CDR2 и CDR3 в вариабельной области легкой цепи (VL) крысиного анти-бычьего PD-L1 антитела 6C11-3A11 (моноклональное антитело), полученного авторами настоящего изобретения, представляют собой область, состоящую из аминокислотной последовательности KSISKY (SEQ ID NO:1), область, состоящую из аминокислотной последовательности SGS, и область, состоящую из аминокислотной последовательности QQHNEYPLT (SEQ ID NO:2), соответственно (см. ФИГ.2).
Кроме того, CDR1, CDR2 и CDR3 в вариабельной области тяжелой цепи (VH) крысиного анти-бычьего PD-L1 антитела 6C11-3A11 представляют собой область, состоящую из аминокислотной последовательности GYTFTDYI (SEQ ID NO:3), область, состоящую из аминокислотной последовательности INPDSGGN (SEQ ID NO:4), и область, состоящую из аминокислотной последовательности ARGITMMVVISHWKFDF (SEQ ID NO:5), соответственно (см. ФИГ.2).
В аминокислотных последовательностях KSISKY (SEQ ID NO:1), SGS и QQHNEYPLT (SEQ ID NO:2), а также в аминокислотных последовательностях GYTFTDYI (SEQ ID NO:3), INPDSGGN (SEQ ID NO:4) и ARGITMMVVISHWKFDF (SEQ ID NO:5) могут быть удалены, замещены или добавлены одна, две, три, четыре или пять аминокислот. Даже если были введены такие мутации, то у полученных аминокислотных последовательностей остается функция антитела против PD-L1 с CDR VL или CDR VH.
Используемый в настоящем документе термин "антитело" представляет собой понятие, охватывающее не только полноразмерные антитела, но и антитела меньших молекулярных размеров, такие как Fab, F(ab)’2, ScFv, диатело, VH, VL, Sc(Fv)2, биспецифический sc(Fv)2, минитело, мономер scFv-Fc и димер scFv-Fc.
Анти-PD-L1 антитело по настоящему изобретению может быть получено у крыс. Например, анти-PD-L1 антитело может быть крысиным анти-бычьим PD-L1 антителом.
Аминокислотная последовательность VL и аминокислотная последовательность VH крысиного анти-бычьего PD-L1 антитела 6С11-3А11 (моноклональное антитело) приведена в SEQ ID NO:6 и 7, соответственно. Аминокислотные последовательности SEQ ID NO:6 и 7 могут иметь делецию(и), замену(ы) или добавление(я) одной или нескольких (например, до пяти, максимум примерно 10) аминокислот. Даже если были введены такие мутации, то у полученных аминокислотных последовательностей остается функция антитела против PD-L1 с VL или VH.
Существует два типа иммуноглобулиновой легкой цепи, которые называются каппа (κ) цепью и лямбда (λ) цепью. В анти-PD-L1 антителе по настоящему изобретению, константная область легкой цепи (CL) может иметь аминокислотную последовательность константной области либо каппа цепи, либо лямбда цепи. При этом, лямбда цепь относительно преобладает у овец, кошек, собак и лошадей, а каппа-цепи преобладает у мышей, крыс, человека и свиней. Крысиное анти-бычье PD-L1 антитело 6C11-3A11 (моноклональное антитело) представляет собой крысиное IgG2a, и его CL имеет аминокислотную последовательность константной области каппа цепи.
Константная область тяжелой цепи (CH) анти-PD-L1 антитела по настоящему изобретению может иметь аминокислотную последовательность константной области крысиного IgG2a. Тяжелая цепь иммуноглобулина классифицируется на цепь γ, цепь μ, цепь α, цепь δ и цепь ε в зависимости от разницы в константной области. В зависимости от типа присутствующей тяжелой цепи образуются пять классов (изотипов) иммуноглобулинов: IgG, IgM, IgA, IgD и IgE.
Иммуноглобулин G (IgG) составляет 70-75% иммуноглобулинов человека и является наиболее часто обнаруживаемым в плазме крови мономерным антителом. IgG имеет структуру из четырех цепей, состоящую из двух легких цепей и двух тяжелых цепей. IgG1, IgG2 и IgG4 человека имеют молекулярную массу примерно 146000, тогда как IgG3 человека имеет длинную шарнирную область, которая соединяет Fab-область и Fc-область и имеет большую молекулярную массу - 170000. IgG1 человека составляет около 65%, IgG2 человека составляет около 25%, IgG3 человека составляет около 7% и IgG4 человека составляет около 3% IgG человека. Они равномерно распределены внутри и снаружи кровеносных сосудов. Обладая сильным сродством к Fc рецепторам и факторам комплемента на поверхности эффекторных клеток, IgG1 человека индуцирует антитело-зависимую клеточную цитотоксичность (ADCC), а также активирует факторы комплемента для индукции комплемент-зависимой клеточной цитотоксичности (CDC). IgG2 и IgG4 человека показывают низкую ADCC и CDC активность, так как у них низкое сродство в отношении Fc рецепторов и факторов комплекта.
Иммуноглобулин М (IgM), на долю которого приходится около 10% иммуноглобулинов человека, представляет собой пентамерное антитело, состоящее из пяти основных четырех-цепочечных структур, соединенных вместе. Его молекулярный вес составляет 970000. Обычно находящийся только в крови, IgM продуцируется в ответ на инфекционные микроорганизмы и отвечает за ранние стадии иммунитета.
Иммуноглобулин А (IgA) составляет 10-15% иммуноглобулинов человека. Его молекулярный вес составляет 160000. Секретируемый IgA представляет собой димерное антитело, состоящее из двух молекул IgA, соединенных вместе. IgA1 содержится в сыворотке крови, выделениях из носа, слюне и грудном молоке. IgA2 в изобилии представлен в желудочном соке.
Иммуноглобулин D (IgD) представляет собой мономерное антитело, на долю которого приходится не более 1% иммуноглобулинов человека. IgD находится на поверхности В-клеток и участвует в стимуляции продукции антител.
Иммуноглобулин Е (IgE) представляет собой мономерное антитело, которое встречается в чрезвычайно малом количестве, составляя всего 0,001% или менее иммуноглобулинов человека. Считается, что иммуноглобулин Е участвует в иммунном ответе на паразитов, но в развитых странах, где паразиты встречаются редко, IgE в значительной степени связан с бронхиальной астмой и аллергией, среди прочего.
Что касается крыс, то последовательности IgG1, IgG2a, IgG2b и IgG2c у них были идентифицированы как тяжелая цепь IgG. Крысиное анти-бычье PD-L1 антитело 6С11-3А11 имеет аминокислотную последовательность CH IgG2a.
В антителе по настоящему изобретению более предпочтительно, чтобы цепь CL имела аминокислотную последовательность константной области каппа и CH имела аминокислотную последовательность константной области IgG2a.
Аминокислотная последовательность и нуклеотидная последовательность VL крысиного анти-бычьего PD-L1 антитела 6С11-3А11, идентифицированные авторами настоящего изобретения, показаны в SEQ ID NO:6 и 14, соответственно.
Аминокислотная последовательность и нуклеотидная последовательность VH крысиного анти-бычьего PD-L1 антитела 6С11-3А11, идентифицированные авторами настоящего изобретения, показаны в SEQ ID NO:7 и 15, соответственно.
Аминокислотная последовательность и нуклеотидная последовательность CL (каппа цепь) крысиного анти-бычьего PD-L1 антитела 6С11-3А11, идентифицированные авторами настоящего изобретения, показаны в SEQ ID NO:8 и 16, соответственно. Эти последовательности идентичны последовательностям, зарегистрированным в GenBank (база данных по нуклеотидным последовательностям, представленная National Center for Biotechnology Information (NCBI)) с номерами доступа #XM_008775358.2, #BC062802.1, #BC088255.1, #L22653.1, #L22655.1 и #M14434.1.
Аминокислотная последовательность и нуклеотидная последовательность CH (IgG2a) крысиного анти-бычьего PD-L1 антитела 6С11-3А11, идентифицированные авторами настоящего изобретения, показаны в SEQ ID NO:9 и 17, соответственно. Эти последовательности идентичны последовательностям, зарегистрированным в GenBank под номером доступа # BC088240. 1, #BC091257.1, #BC091272.1, #BC088423.1, #L22652.1 и #L22654.1.
Аминокислотные последовательности и нуклеотидные последовательности CL и CH для крысиных антител, отличные от указанных выше, могут быть получены из известных баз данных для использования в настоящем изобретении.
В качестве аминокислотной последовательности и нуклеотидной последовательности каппа цепи Ig крысы последовательность, зарегистрированная в GenBank под регистрационным номером #V01241.1 показана в SEQ ID NO:10 и 18.
В качестве аминокислотной последовательности и нуклеотидной последовательности каппа цепи Ig крысы последовательность, зарегистрированная в GenBank под регистрационным номером #X16129.1, показана в SEQ ID NO:11 и 19.
В качестве аминокислотной последовательности и нуклеотидной последовательности каппа цепи Ig крысы последовательность, зарегистрированная в GenBank под регистрационным номером #DQ402471.1, показана в SEQ ID NO:12 и 20.
В качестве CH IgG2a крысы, последовательность, зарегистрированная в Genbank под регистрационным номером #DQ402472. 1, показана в SEQ ID NO:13 и 21.
Анти-PD-L1 антитело по настоящему изобретению может представлять собой анти-PD-L1 антителу, в котором CL имеет аминокислотную последовательность с любой SEQ ID NO:8 и 10-12, и CH имеет аминокислотную последовательность SEQ ID NO:9 или 13.
Аминокислотные последовательности SEQ ID NO:8-13 могут иметь делецию(и), замену(ы) или добавление(я) одной или нескольких (например, до пяти, максимум примерно 10) аминокислот. Даже если были введены такие мутации, то у полученных аминокислотных последовательностей остается функция антитела против PD-L1 с CL или CH.
Выравнивание аминокислотных последовательностей CL и CH крысиного анти-PD-L1 антитела показаны на ФИГ.8 и ФИГ.9, соответственно. Вышеописанные мутации, такие как делеция, замена или добавление аминокислот, могут соответствующим образом происходить на сайтах мутации как показано на ФИГ.8 и 9 или в непосредственной близости от них.
Анти-PD-L1 антитело по настоящему изобретению может быть химерным антителом. VL и VH антитела могут быть соответственно получену у крыс. Например, VL может быть VL крысиного анти-PD-L1 антитела (например, 6C11-3A11); VH может быть VH крысиного анти-PD-L1 антитела; и CL и CH могут быть получены у животного, отличного от крысы. Например, если антитело крысы химеризуется с помощью константных областей антитела мыши, то полученное химерное антитело может использоваться для тестирования и диагностики, поскольку различные вторичные антитела к антителам мыши коммерчески доступны. Аминокислотные последовательности и нуклеотидные последовательности CL и CH антител животных, отличных от крыс, могут быть получены из известных баз данных для использования в настоящем изобретении.
Аминокислотные последовательности и нуклеотидные последовательности CL и CH для человека, мыши, крупного рогатого скота, собак, овец, свиней и буйволов приведены в таблице ниже.
Таблица

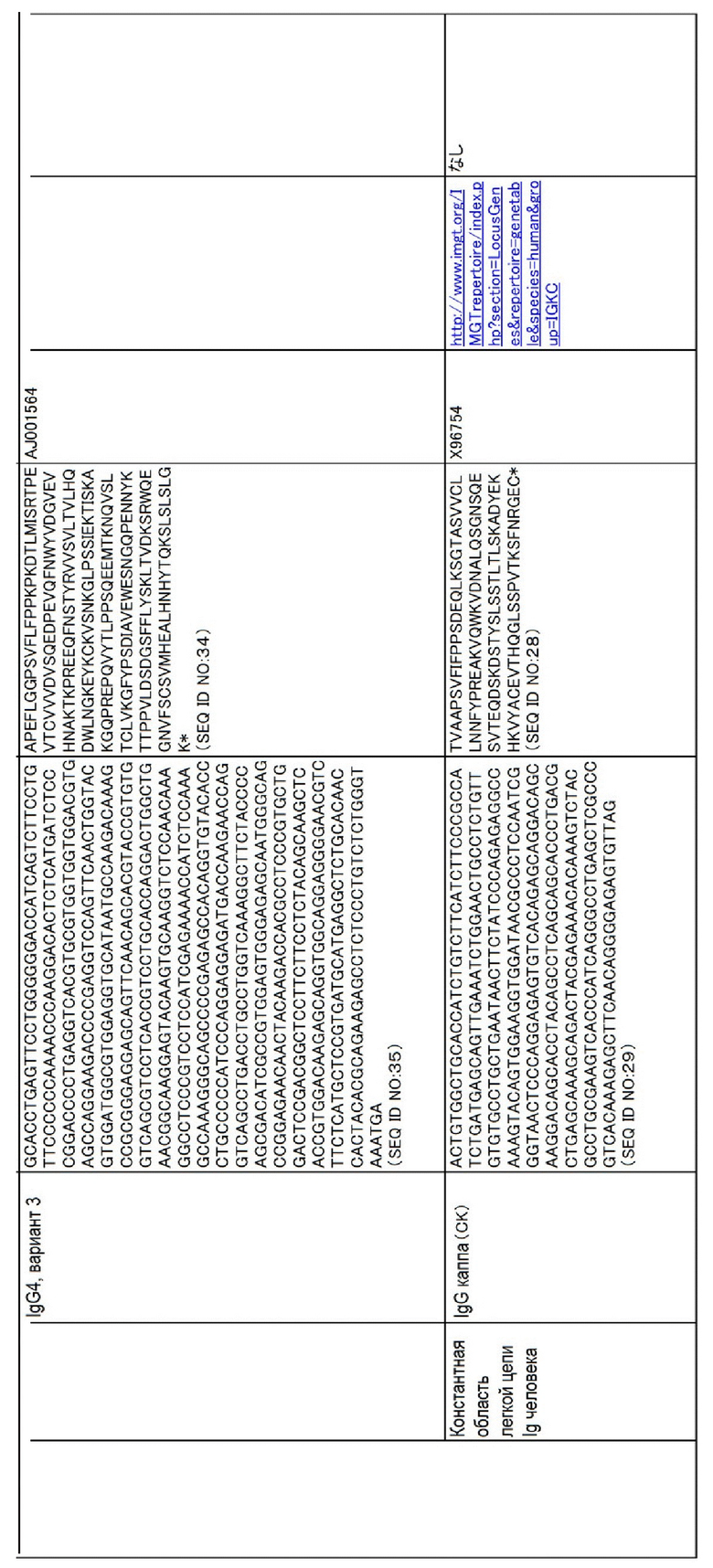
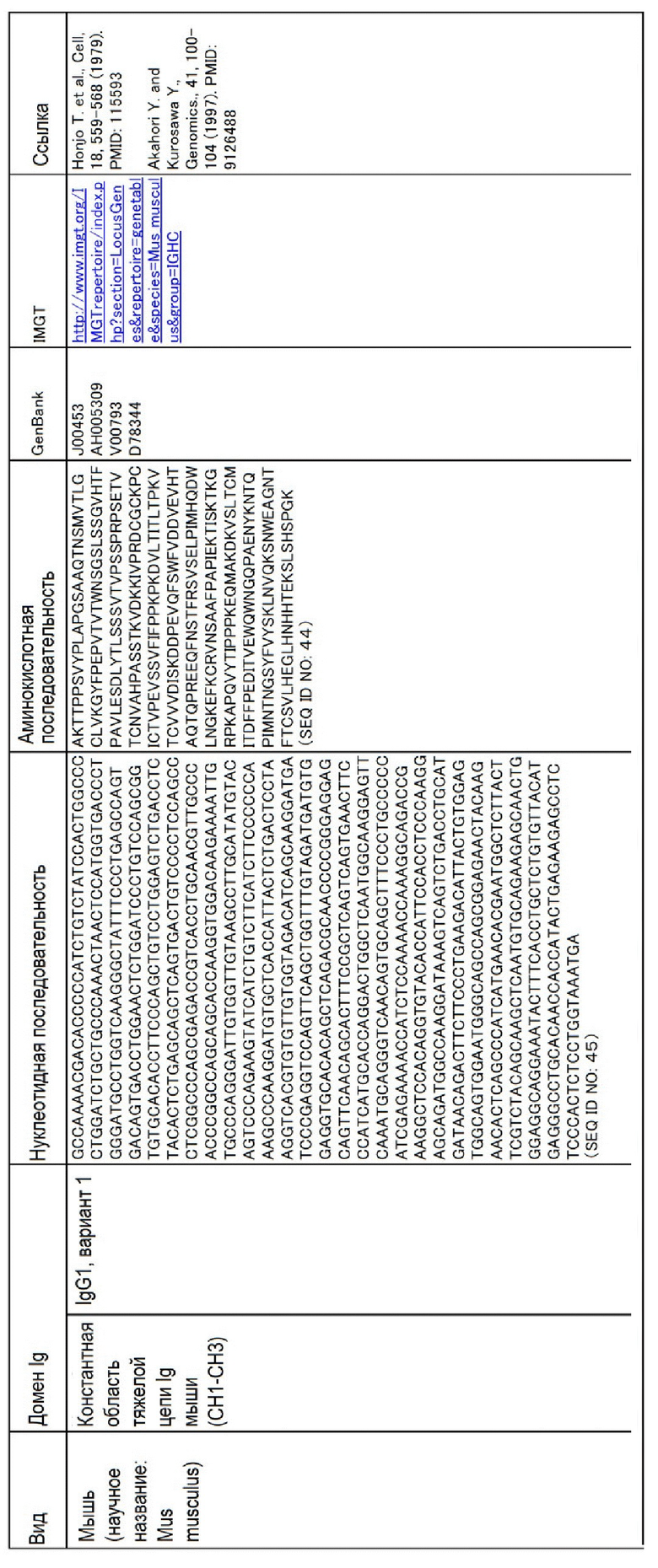
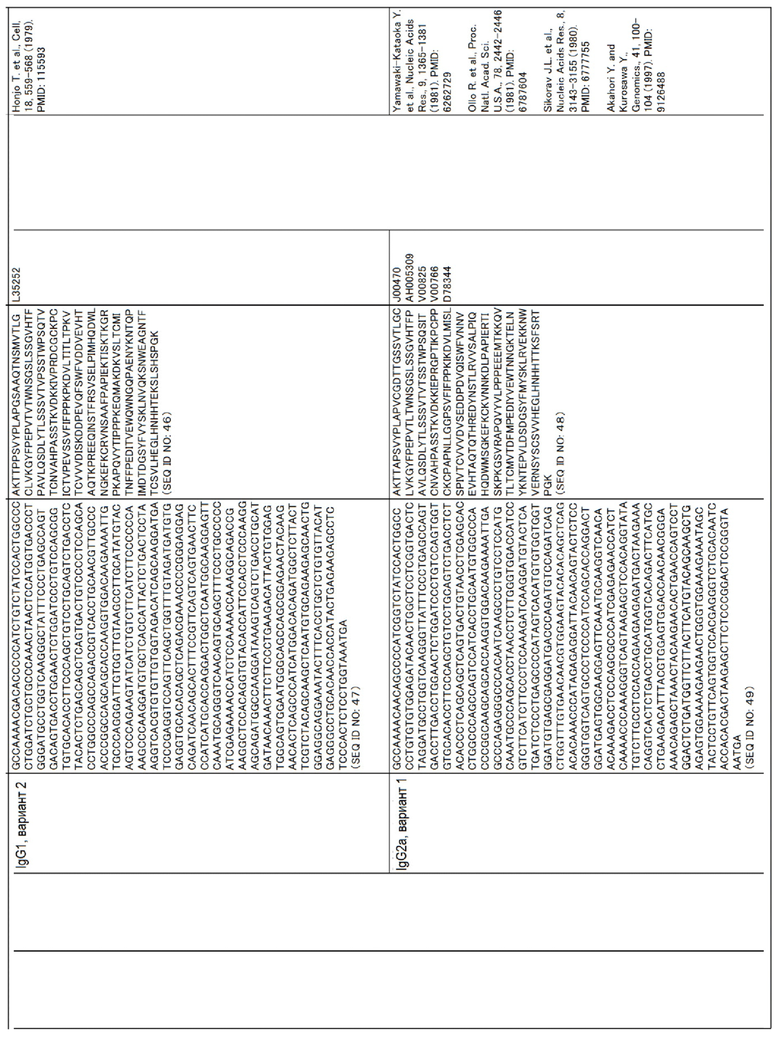
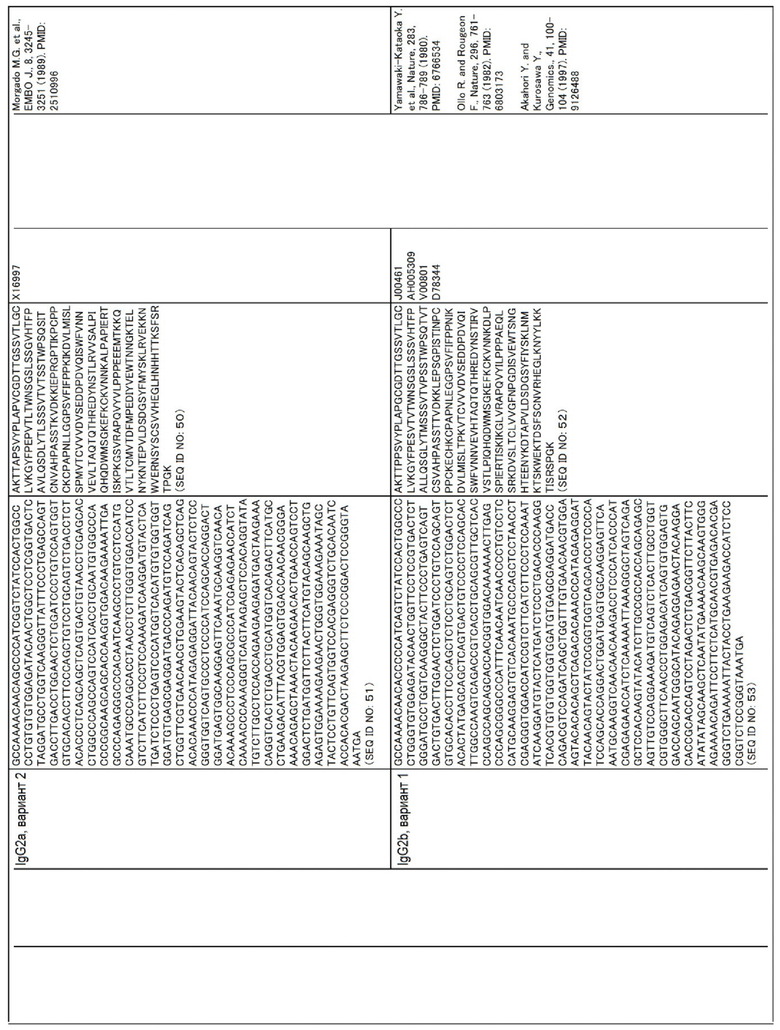

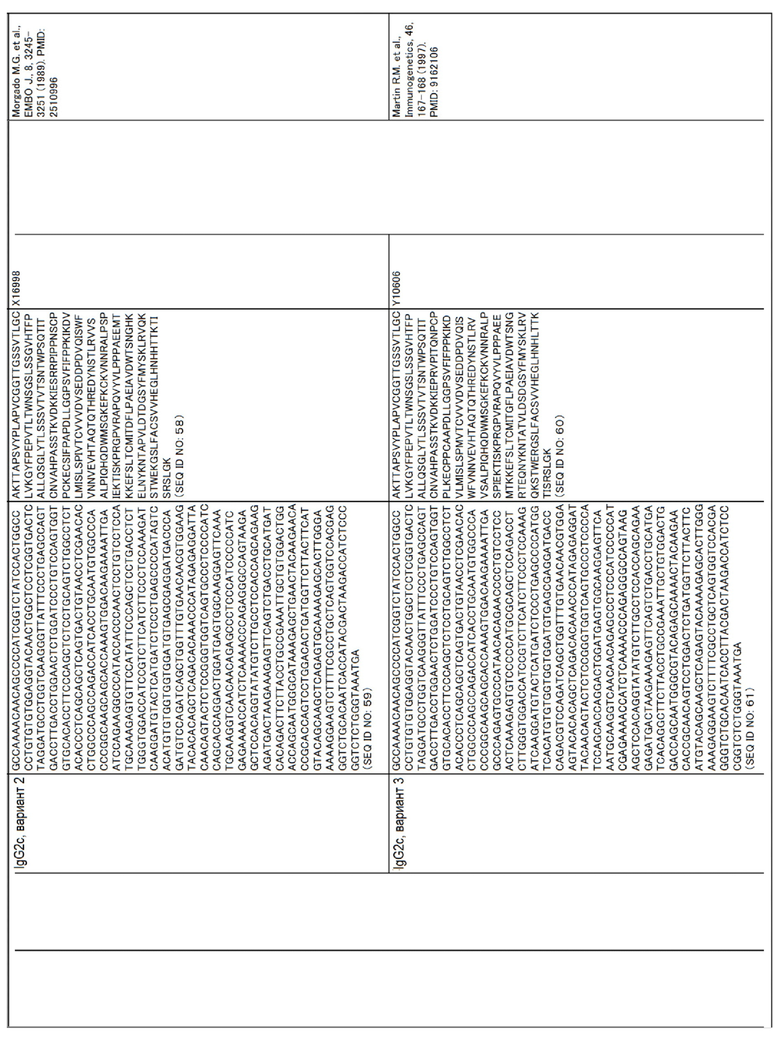

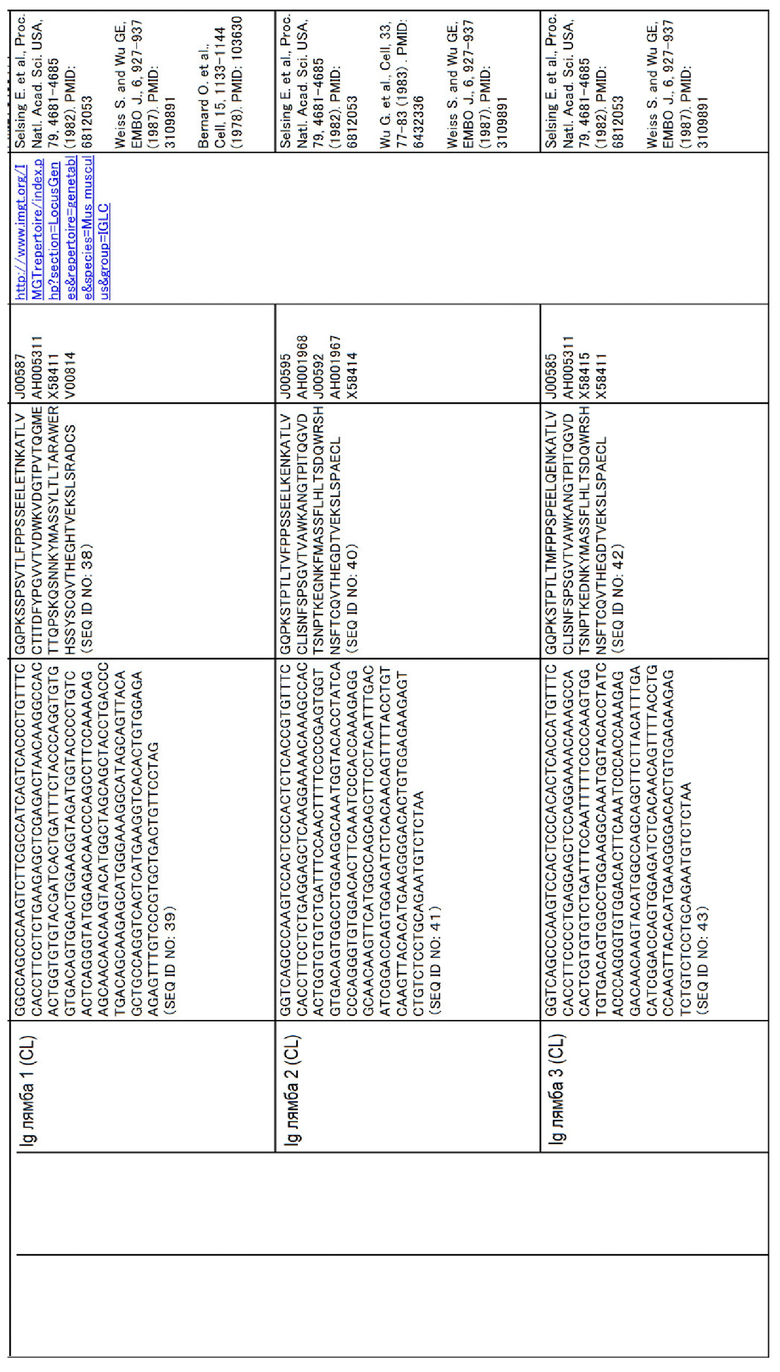


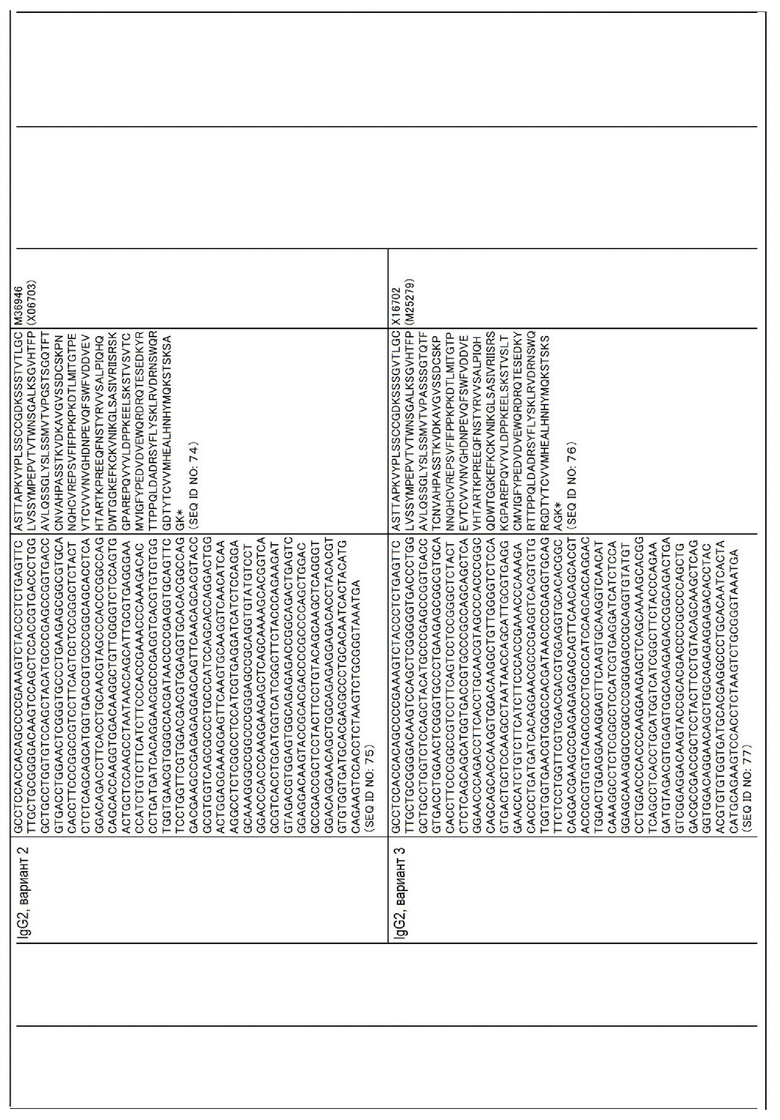
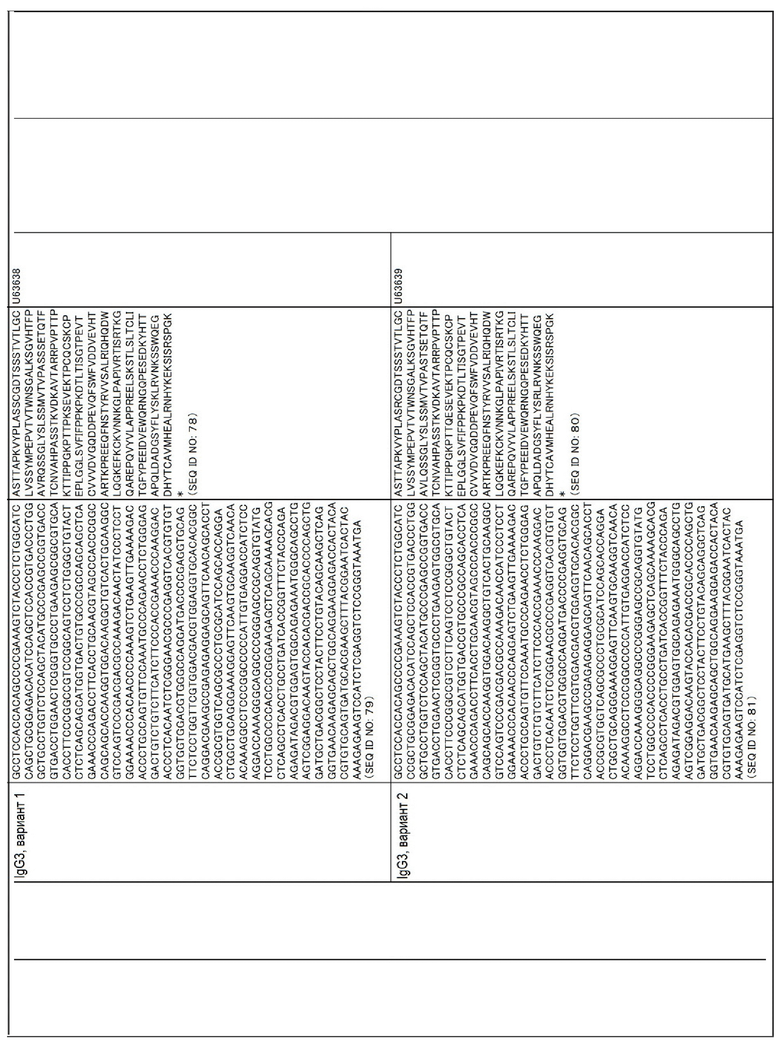









Аминокислотные последовательности SEQ ID NO:8-13, 28, 30, 32, 34, 36, 38, 40, 42, 44, 46, 48, 50, 52, 54, 56, 58, 60, 62, 64, 66, 68, 70, 72, 74, 76, 78, 80, 82, 84, 86, 88, 90, 92, 94, 96, 98, 100, 102, 104, 106, 108, 110, 112, 114, 116, 118, 120 и 122 могут иметь делецию(и), замену(ы) или добавление(я) одной или нескольких(например, до пяти, максимум примерно 10) аминокислот. Даже если были введены такие мутации, то у полученных аминокислотных последовательностей остается функция как у константной области тяжелой или легкой цепи Ig.
Анти-PD-L1 антитело по настоящему изобретению может иметь четырехцепочечную структуру, содержащую две легкие цепи и две тяжелые цепи.
Анти-PD-L1 антитело по настоящему изобретению может быть получено как описано ниже. Коротко, синтезируют искусственный ген, который содержит последовательность легкой цепи (последовательность вариабельной области и последовательность константой области) и последовательность тяжелой цепи (последовательность вариабельной области и последовательность константной области) анти-PD-L1 антитела по настоящему изобретению. Полученный ген встраивают в вектор (например, плазмиду), который затем вводят в клетку-хозяина (например, клетку млекопитающего, такую как клетка CHO). Клетку-хозяина культивируют, и интересующее антитело собирают из полученной культуры. При синтезе искусственного гена могут быть оптимизированы кодоны нуклеотидной последовательности.
Настоящее изобретение относится к ДНК, кодирующей анти-PD-L1 антитело, содержащее (a) легкую цепь, содержащую CDR1 с аминокислотной последовательностью KSISKY (SEQ ID NO:1), CDR2 с аминокислотной последовательностью SGS, и CDR3 с аминокислотной последовательностью QQHNEYPLT (SEQ ID NO:2), и (b) тяжелую цепь, содержащую CDR1 с аминокислотной последовательностью GYTFTDYI (SEQ ID NO:3), CDR2 с аминокислотной последовательностью INPDSGGN (SEQ ID NO:4), и CDR3 с аминокислотной последовательностью ARGITMMVVISHWKFDF (SEQ ID NO:5). Настоящее изобретение также относится к ДНК, кодирующей легкую цепь анти-PD-L1 антитела, содержащей CDR1 c аминокислотной последовательностью KSISKY (SEQ ID NO:1), CDR2 с аминокислотной последовательностью SGS, and CDR3 с аминокислотной последовательностью QQHNEYPLT (SEQ ID NO:2) (ДНК по (а’)). Кроме того, настоящее изобретение относится к ДНК, кодирующей тяжелую цепь анти-PD-L1 антитело, содежращей CDR1 с аминокислотной последовательностью GYTFTDYI (SEQ ID NO:3), CDR2 с аминокислотной последовательностью INPDSGGN (SEQ ID NO:4), и CDR3 с аминокислотной последовательностью ARGITMMVVISHWKFDF (SEQ ID NO:5) (ДНК по (b’)).
В случае (а) легкая цепь, содержащая CDR1 с аминокислотной последовательностью KSISKY (SEQ ID NO:1), CDR2 с аминокислотной последовательностью SGS, и CDR3 с аминокислотной последовательностью QQHNEYPLT (SEQ ID NO:2), и (b) тяжелая цепь, содержащая CDR1 с аминокислотной последовательностью GYTFTDYI (SEQ ID NO:3), CDR2 с аминокислотной последовательностью INPDSGGN (SEQ ID NO:4), и CDR3 с аминокислотной последовательностью ARGITMMVVISHWKFDF (SEQ ID NO:5), должна быть сделана ссылка на приведенное выше описание. ДНК, содержащая ДНК (a‘) и ДНК ('b), может быть синтезирована на коммерческом синтезаторе. К этой ДНК могут быть добавлены сайты распознавания ферментов рестрикции, последовательности Козака, последовательности поли-А дополнительного сигнала, промоторные последовательности, интронные последовательности или тому подобное.
Настоящее изобретение также относится к вектору, содержащему указанную выше ДНК, кодирующую анти-PD-L1 антитело.
В качестве вектора можно использовать плазмиды на основе Escherichia coli (например, pBR322, pBR325, pUC12 или pUC13); плазмиды на основе Bacillus subtilis (например, pUB110, pTP5 или pC194), плазмиды на основе дрожжей (например, pSH19 или pSH15); бактериофаги, например фаг λ; вирусы животных, такие как ретровирусы или вирус осповакцины; или вирусы на основе патогенов насекомых, такие как бакуловирус. В примерах, описанных ниже, используется pDC6 (японский патент № 5704753, патент США 9096878, патент ЕС 2385115, патент Гонконга (Китай), патент HK1163739 и патент Австралии 2009331326).
Вектор может также содержать промоторы, усилители, сигналы сплайсинга, поли-А дополнительные сигналы, последовательности интронов, маркеры селекции, точки начала репликации SV40 и тому подобное.
Настоящее изобретение также относится к клетке-хозяину, трансформированной вышеуказанным вектором. Можно получить анти-PD-L1 антитело по настоящему изобретению путем культивирования клетки-хозяина и сбора интересующего антитела из из полученной культуры. Поэтому, настоящее изобретение также относится к способу получения антитела, включающему культивирование вышеописанной клетки-хозяина и сбор анти-PD-L1 антитела по изобретению из культуры. В способе по настоящему изобретению для получения антитела вектор, содержащий ДНК, содержащую ДНК, кодирующую легкую цепь, и ДНК, кодирующую тяжелую цепь, может быть трансфицирован в клетку-хозяин. Альтернативно, вектор, в который встроена ДНК, кодирующая легкую цепь, и вектор, в который встроена ДНК, кодирующая тяжелую цепь, могут быть совместно трансфицированы в клетку-хозяин.
Примеры клеток-хозяев включают, но не ограничиваются ими, бактериальные клетки (например, бактерия Escherichia, бактерия Bacillus или Bacillus subtilis), клетки грибов (например, дрожжи или Aspergillus), клетки насекомых (например, клетки S2 или клетки Sf), животные клетки (такие как клетки CHO, клетки COS, клетки HeLa, клетки C127, клетки 3T3, клетки BHK или клетки HEK 293) и клетки растений. Среди этих клеток предпочтительными являются клетки CHO-DG44 (CHO-DG44(dfhr-/-)), которые являются клетками с дефицитом дигидрофолатредуктазы.
Введение рекомбинантного вектора в клетку-хозяин может быть осуществлено способами, описанными в Molecular Cloning 2nd Edition, J. Sambrook et al., Cold Spring Harbor Lab. Press, 1989 (например, способом с фосфатом кальция, способом с DEAE-декстраном, трансфекцией, микроинъекцией, липофекцией, электропорацией, трансдукцией, введением при соскабливании, метод "дробовика" и тому подобное) или инъекцией.
Полученный трансформант можно культивировать в среде с последующим сбором анти-PD-L1 антитела по настоящему изобретению из культуры. Если антитело секретируется в среду, то среда может быть восстановлена с последующим выделением и очисткой антитела из среды. Если антитело продуцируется внутри трансформированных клеток, то клетки могут быть лизированы с последующим выделением и очисткой антитела от клеточного лизата.
Примеры среды включают, но не ограничиваются ими, среду OptiCHO, среду Dynamis, среду CD CHO, среду ActiCHO, chtle FortiCHO, среду Ex-Cell CD CHO, среду BalanCD CHO, среду ProCHO 5 и среду Cellvento CHO-100.
рН среды изменяется в зависимости от культивируемой клетки. Как правило, используется диапазон рН от 6,8 до 7,6; в основном, подходящим является диапазон рН от 7,0 до 7,4.
Если клетка, подлежащая культивированию, является клеткой СНО, то культивирование можно проводить способами, известными специалистам в данной области. Например, обычно можно проводить культивирование в газофазной атмосфере с концентрацией CO2, равной 0-40%, предпочтительно 2-10%, при 30-39°С, предпочтительно при примерно 37°C.
Соответствующий период культивирования обычно составляет от одного дня до трех месяцев, предпочтительно от одного дня до трех недель.
Выделение и очистку антитела можно проводить известными способами. Известные способы выделения/очистки, которые могут быть использованы в настоящем изобретении, включают, но не ограничиваются ими, способы, основанные на различной растворимости (такие как высаливание или осаждение); способы на основе разницы в молекулярной массе (такие как диализ, ультрафильтрация, гель-фильтрация или SDS-электрофорез в полиакриламидном геле); способы на основе разницы в электрических зарядов (такие как, ионообменная хроматография); способы на основе специфичности сродства (такие как, аффинная хроматография); способы на основе различие в гидрофобности (такие как, высокоэффективная жидкостная хроматография с обращенной фазой); и способы на основе разницы в изоэлектрической точки (такие как изоэлектрическое фокусирование).
Также можно получать анти-PD-L1 антитела по настоящему изобретению путем культивирования гибридомы, которая может быть получена способом, описанным в литературе (Ikebuchi R, Konnai S, Okagawa T, Yokoyama K, Nakajima C, Suzuki Y, Murata S, Ohashi K, Immunology. 2014 Aug;142(4):551-61). Гибридома, продуцирующая анти-PD-L1 антитело 6C11-3A11, хранится в лаборатории автором настоящего изобретения (Laboratory of Infectious Diseases, Department of Disease Control, Faculty of Veterinary Medicine, Hokkaido University).
Анти-PD-L1 антитело по настоящему изобретению можно использоваться для детекции PD-1. Таким образом, настоящее изобретение относится к композиции для детекции PD-L1, содержащей анти-PD-L1 антитело в качестве активного ингредиента.
Детекция PD-L1 может быть осуществлена такими способами, включая, но не ограничиваясь ими, как иммуногистохимическое окрашивание, иммуноцитохимическое окрашивание, проточная цитометрия, иммуноферментный анализ (ELISA) и Вестерн-блоттинг.
Аналиты для детекции могут быть представлены образцами, такими как ткани или жидкости организма, взятые из организмов (например, кровь (цельная кровь, плазма, сыворотка или специфические клетки, такие как эритроциты, лейкоциты или лимфоциты), моча, слюна и тому подобное); культура клеток; и культивируемые клетки (разработанные клеточные линии, первичные культивируемые клетки, субкультурные клетки и тому подобное). Источник таких аналитов конкретно не ограничен. Примеры включают крысу, собаку, овцу, козу, свинью, кошку, человека, лошадь, крупный рогатый скот, буйвола, яка, кролика, мышь, хомяка и морскую свинку.
Антитело против PD-L1 по настоящему изобретению может быть мечено радиоизотопами, ферментами, люминесцентными веществами, флуоресцентными веществами, биотином или тому подобное. Если за реакцией с первым антителом (анти-PD-L1 антитело по настоящему изобретению), которое специфически связывается с молекулой-мишенью (PD-L1), следует реакция со вторым антителом, которое связывается с первым антителом для детекции молекулы-мишени, то целесообразно метить второе антитело.
Поскольку PD-L1 экспрессируется в раковых клетках и вирусно-инфицированных клетках на высоком уровне, то композиция по настоящему изобретению может быть использована для диагностики злокачественных новообразований и/или инфекций. Обычно количество (концентрация) PD-L1 в аналите определяется на основе количества (концентрации) комплекса PD-L1 и анти-PD-L1 антитела. Если количество (концентрация) PD-L1 в аналите выше, чем в отрицательном контроле (например, в здоровой окружающей ткани (соединительная ткань, кровеносные сосуды и тому подобное)), то аналит может быть диагностирован как пораженный злокачественным новообразованием и/или инфекцией. Альтернативно, если в аналите детектирван PD-L1, то аналит может быть диагностирован как пораженный злокачественным новообразованием и/или инфекцией.
Примеры злокачественных новообразований и/или инфекций включают, но не ограничиваются ими, неопластические заболевания (например, злокачественная меланома, рак легких, рак желудка, рак почки, рак молочной железы, рак мочевого пузыря, рак пищевода, рак яичников и тому подобное), лейкоз, болезнь Джона, анаплазмоз, бактериальный мастит, грибковый мастит, инфекции, вызванные микоплазмой (например, мастит, вызванный микоплазмой, пневмония, вызванная микоплазмой, или тому подобное), туберкулез, инфекцию, вызванную Theileria orientalis, криптоспоридоз, кокцидиоз, трипаносомоз и лейшманиоз.
Композиция по настоящему изобретению может использоваться для селекции подопытных животных, подходящих для терапии на основе анти-PD-L1 антитела. Например, животные, удовлетворяющие следующим двум критериям, могут рассматриваться как животные-кандидаты.
1. В случае диагностирования при медико-патологическом исследовании злокачественного новообразования (например меланомы) или инфекции
2. В случае, считающемся положительным для анти-PD-L1 антитела
Отрицательным контролем может быть здоровая окружающая ткань (соединительная ткань, кровеносные сосуды и тому подобное), а положительным контролем может быть случай злокачественного новообразования (такой как меланома) или инфекции. В основном клинические испытания можно проводить с животными с опухолью, которая является положительной на иммуногистохимическое окрашивание практически во всех областях.
Такие животными являются, но не ограничиваясь ими, крыса, собака, овца, коза, свинья, кошка, человек, лошадь, крупный рогатый скот, буйвол, як, кролик, мышь, хомяк и морская свинка.
Композиция по настоящему изобретению может дополнительно содержать реагенты для детекции меток, разбавители, жидкости для промывки, инструкции, описывающие критерии диагностики/селекции, и тому подобное.
Примеры
Далее, настоящее изобретение описано более подробно со ссылкой на Примеры. Однако, настоящее изобретение не ограничивается этими Примерами.
[Пример 1]
1. Введение
Белок запрограммированной клеточной гибели 1 (PD-1), являющийся иммуноингибиторным рецептором, и лиганд запрограмированной клеточной гибели 1 (PD-L1) являются молекулами, идентифицированными профессором Tasuku Honjo et al., Kyoto University, как факторы, которые ингибируют чрезмерный иммунный ответ и которые тесно связаны с иммунотолерантностью. Недавно было выяснено, что эти молекулы также участвуют в иммуносупрессии при инфекциях и опухолях у различных животных. В рассматриваемом Примере анти-бычье PD-L1 моноклональное антитело получали путем иммунизации крыс, а затем отбирали клон (6Cc11-3А11), способный детектировать PD-L1 собаки. Кроме того, авторы настоящего изобретения провели иммуногистохимическое окрашивание для установления эффективности этого анти-бычьего антитела PD-L1 6C11-3A11 при детекции PD-L1 в злокачественных опухолях собаки (таких как меланома) и инфекциях свиней/овец.
2. Материалы и методы
2.1 Клетки, продуцирующие крысиное анти-бычье PD-L1 моноклональное антитело
Определяли нуклеотидную последовательность бычьего PD-L1 (Ikebuchi R, Konnai S, Shirai T, Sunden Y, Murata S, Onuma M, Ohashi K. Vet Res. 2011 Sep 26;42:103). На основе информации о последовательности получали рекомбинантный бычий PD-L1. Крысу иммунизировали в подушечку стопы этим рекомбинантным белком, и получали гибридомы методом с использованием подвздошных лимфатических узлов. В результате, были получены гибридомы, продуцирующие крысиные анти-бычьи PD-L1 моноклональные антитела (Ikebuchi R, Konnai S, Okagawa T, Yokoyama K, Nakajima C, Suzuki Y, Murata S, Ohashi K. Immunology 2014 Aug.; 142(4):551-561). Крысиное анти-бычье PD-L1 антитело 6С11-3А11 является одним из моноклональных антител, полученных от вышеописанной иммунизированной крысы.
2.2 Идентификация полноразмерного гена PD-L1 собаки
Для определения полноразмоной кДНК PD-L1 собаки, сначала конструировали ПЦР-праймеры на основе предполагаемой нуклеотидной последовательности PD-L1 собаки, уже зарегистрированной в The National Center for Biotechnology Information (NCBI) (номер доступа в GenBank; XM_541302). Вкратце, конструировали праймеры для амплификации внутренней последовательности открытой рамки считывания (ORF) этого гена (cPD-1 внутренний F и R), и проводили ПЦР. Для амплифицированных продуктов нуклеотидные последовательности определяли с помощью капиллярного секвенсора согласно общепринятым методам. Затем, для определения нуклеотидной последовательности полноразмерной кДНК PD-L1 констрировали праймеры (cPD-L1 5' GSP и 3' GSP) на основе последовательностей кДНК PD-L1 собами, как определено выше. Осуществляли 5'-RACE и 3'-RACE, используя, соответственно, систему 5′-RACE для быстрой амплификации концов кДНК и систему 3'-RACE для быстрой амплификации концов кДНК (Invitrogen). Полученные фрагменты гена секвенировали, как описано выше (Maekawa N, Konnai S, Ikebuchi R, Okagawa T, Adachi M, Takagi S, Kagawa Y, Nakajima C, Suzuki Y, Murata S, Ohashi K. PLoS One. 2014 Jun 10;9(6):e98415).
Праймер (cPD-L1 внутренний F): ATGAGAATGTTTAGTGTCTT (SEQ ID NO:22)
Праймер (cPD-L1 внутренний R): TTATGTCTCTTCAAATTGTATATC (SEQ ID NO:23)
Праймер (cPD-L1 5'GSP): TTTTAGACAGAAAGTGA (SEQ ID NO:24)
Праймер (cPD-L1 3'GSP): GACCAGCTCTTCTTGGGGAA (SEQ ID NO:25)
2.3 Получение клеток COS-7, экспрессирующих PD-L1 собаки
Для получения плазмиды экспрессии PD-L1-EGFP собаки, осуществляли ПЦР, используя в качестве матрицы синтезированную кДНК из PBMC бигля и праймеры, сконструированные путем добавления сайтов распознавания BglII и EcoRI на 5'-конец (cPD-L1-EGFP F и R). Полученные продукты ПЦР расщепляли BglII (New England Biolabs) и EcoRI (Takara), а затем очищали, используя набор Fastgene Gel/ПЦР Extraction Kit (NIPPON Genetics), и затем клонировали в вектор pEGFP-N2 (Clontech), обработанный аналогичным образом ферментами рестрикции. Полученную интересующую плазмиду экспрессии экстрагировали, используя набор QIAGEN Plasmid Midi kit (Qiagen), и хранили при -30°C перед использованием в экспериментах. Далее, полученная таким образом плазмида экспрессии обозначена как pEGFP-N2-cPD-L1.
Праймер (cPD-L1-EGFP F): GAAGATCTATGAGAATGTTTAGTGTC (SEQ ID NO:26)
Праймер (cPD-L1-EGFP R): GGAATTCTGTCTCTTCAAATTGTATATC (SEQ ID NO:27)
Клетки COS-7 субкультивировали при плотности 5х104 клеток/см2 в 6-и луночных планшетах, а затем культивировали в течение ночи в среде RPMI 1640, содержащей 10% инактивированной фетальной бычьей сыворотки и 0,01% L-глютамина при 37°C в присутствии 5% CO2. PEGFP-N2-cPD-L1 или pEGFP-N2 (отрицательный контроль) вводили в клетки COS-7 в количестве 0,4 мкг/см2, используя Lipofectamine 2000 (Invitrogen). Клетки культивировали в течение 48 часов (клетка, экспрессирующая cPD-L1-EGFP собаки, и клетка, экспрессирующая EGFP). Для подтверждения экспрессии PD-L1 в полученных таким образом экспрессирующих клетках, визуализировали внутриклеточную локализацию усиленного зеленого флуоресцентного белка (EGFP) с помощью инфертированного конфокального лазерного микроскопа LSM700 (ZEISS) (Maekawa N, Konnai S, Ikebuchi R, Okagawa T, Adachi M, Takagi S, Kagawa Y, Nakajima C, Suzuki Y, Murata S, Ohashi K. PLoS One. 2014 Jun 10;9(6):e98415).
2.4 Перекрестное взаимодействие крысиного анти-бычьего PD-L1 антитела 6С11-3А11 с PD-L1 собаки
Для того, чтобы подтвердить, что крысиное анти-бычье PD-L1 антитело 6C11-3A11 специфически связывается с PD-L1 собаки, осуществляли проточную цитометрию с клетками, экспрессирующими cPD-L1-EGFP, или клетками, эксперссирующими EGFP, полученными в разделе 2.3 выше. К клетками в количестве 2×105-1×106 добавляли 10 мкг/мл анти-бычьего антитела PD-L1 6C11-3A11 и проводили реакцию в течение 30 мин при комнатной температуре. После промывки осуществляли дектецию связывания антител на клеточной поверхности с помощью козлиного анти-крысиного Ig антитела, меченного аллофикоцианином (Beckman Coulter). Для анализа использовали FACS Verse (Becton, Dickinson and Company). В качестве отрицательного контрольного антитела использовали изотипический контроль IgG2a (κ) крысы (BD Bioscience). Для каждой операции промывки и разведения антител использовали 10% инактивированную козью сыворотку с добавлением PBS. Результаты приведены на фиг.1.
2.5 Анализ CDR крысиного анти-бычьего PD-L1 антитела 6Cc11-3А11
Из гибридомы, продуцирующей антитело, определяли гены тяжелой цепи и легкой цепи крысиного анти-бычьего PD-L1 антитела 6С11-3А11 из гибридомы, используя метод RACE. Определяющие комплементарность области (CDR) крысиного анти-бычьего PD-L1 антитела 6Cc11-3А11 определяли с помощью NCBI IGBLAST (http://www.ncbi.nlm.nih.gov/igblast/). Результаты приведены на Фиг.2.
2.6 Иммуногистохимическое окрашивание опухолевых тканей собак и инфицированных тканей овец/свиней
Для подтверждения того, что крысиное анти-бычье антитело против PD-L1 6C11-3A11 можно использовать для иммуногистохимического окрашивания PD-L1 опухолей у собак, образцы опухолей, фиксированные формалином в парафине, иммуногистохимически окрашивали. Согласно общепринятым методам полученные образцы депарафинизировали и затем подвергали микроволновой обработке (5 минут, дважды) в цитратном буфере. Затем образцы подвергали воздействию антителом PD-L1 6C11-3A11 (400-кратное разведение) в течение 30 минут, а затем с Simple Stain Mouse MAX-PO (крыса) (Nichirei Bioscience) в течение 30 минут. Для окрашивания обрабатывали диаминобензидином (DAB) в течение 10 минут.
Результаты показаны на Фиг. 3, 4, 5-1, 5-2, 6 и 7.
Анти-MelamA антитело, единственное коммерчески доступное антитело, специфичное к меланоме, окрашивало опухолевые клетки очень плохо (Фиг.3, слева). С другой стороны, анти-PD-L1 антитело (6C11-3A11), полученное авторами настоящего изобретения, окрашивало опухолевые клетки очень хорошо (Фиг.3, справа). Анти-PD-L1 антитело (6C11-3A11) было способно окрашивать меланому почти в каждом случае.
При меланоме собак было обнаружено, что опухолевые клетки являются диффузно положительными для анти-PD-L1 антитела (6C11-3A11). (Положительное число/тестированное число=12/12; уровень положительного результата 100%)
При лимфоме собак было обнаружено, что опухолевые клетки являются диффузно положительными для анти-PD-L1 антитела (6C11-3A11). При остеосаркоме собак некоторые опухолевые клетки окрашивались внутриклеточно. При почечно-клеточном раке собак было обнаружено, что опухолевые клетки являются диффузно положительными в различных типах тканей.
В случае листериоза овец изображение с окрашиванием PD-L1 головного мозга при листериозе овец с неврологическими симптомами показано на Фиг.6, левая панель. На увеличенной фотографии этого изображения экспрессия PD-L1 отмечалась в макрофагах, инфильтрирующих очаги поражения головного мозга (Фиг.6, справа).
В случае цирковирусной инфекции свиней типа 2 PD-L1 окрашивался в лимфоидных фолликулах, и вирус окрашивался в этих клетках (Фиг.7, слева).
В случае микоплазменной пневмонии свиней, большое количество макрофагов инфильтрировали поражения в легких, и PD-L1 окрашивались в эти инфильтрирующих клетках (Фиг.7, справа).
Как описано выше, анти-бычье PD-L1 антитело 6C11-3A11 можно использовать для детекции PD-L1 в различных опухолях собак (таких как злокачественная меланома) и инфекциях овец/свиней при иммуногистохимическом окрашивании. Это говорит о возможности использовать анти-бычье PD-L1 антитело 6C11-3A11 для диагностики различных животных и различных заболеваний.
[Пример 2]
1. Введение
Моноклональные антитела могут быть получены путем культивирования гибридом и очистки антител из полученных культур супернатантов. Альтернативно, если определена нуклеотидная последовательность интересующего антитела, то клетка, экспрессирующая антитело, может быть получена путем трансфекции культивированных клеток вектором для экспрессии нуклеотидной последовательности; полученная таким образом клетка, экспрессирующая антитело, может быть использована в качестве замены гибридомы. В рассматриваемом ниже примере будет приведен пример способа, в котором антитело продуцируется системой для экспрессии белка, используя вектор экспрессии и клетки млекопитающего.
2. Материалы и методы
2.1 Получение вектора экспрессии крысиного анти-бычьего антитела PD-L1 6C11-3A11
На основе нуклеотидной последовательности крысиного анти-бычьего PD-L1 антитела 6C11-3A11, определенной в разделе 2.5 Примера 1 выше, осуществляли синтез гена так, чтобы сайт распознавания фермента рестрикции NotI, последовательность Козака, последовательность легкой цепи антитела, последовательность поли-А дополнительного сигнала (PABGH), последовательность промотора (PCMV), сайт распознавания фермента рестрикции SacI, последовательность интрона (INRBG), последовательность Козака, последовательности тяжелой цепи антитела, сайт распознасвания фермента рестикции XbaI были расположены в приведенном порядке. В этом случае кодоны гена антитела могут быть оптимизированы заранее в зависимости от типа клетки, которая должна экспрессировать ген. Цепь синтезированного гена встраивают в вектор экспрессии pDC6 (любезно предоставлен профессором S. Suzuki, Rsearch Center for Zoonosis Control, Hokkaido University) в сайт клонирования (сайты распознавания ферментов рестрикции NotI и XbaI расположены после PCMV и между INRBG и PABGH), используя сайты распознавания ферментов рестрикции так, что перечисленные выше последовательности будет находиться в указанном выше порядке с получением, тем самым, вектора pDC6, экспрессирующего крысиное анти-бычье PD-L1 антитело.
2.2 Экспрессия крысиного анти-бычьего PD-L1 антитела 6C11-3A11
Вектор, экспрессирующий крысиное анти-бычье антитело PD-L1, pDC6, полученный в разделе 2.1 выше, трансфицировали в клетки CHO-DG44 (CHO-DG44(dfhr-/-)), которые являются клетками с дефицитом дигидрофолатредуктазы, и клоны с высокой экспрессией отбирали dot-блоттингом. Для повышения экспрессии амплификацию генов можно выполнять путем добавления нагрузки на клетки в среде, содержащей 60 нМ, 250 нМ или 1000 нМ метотрексата (Mtx). Полученные таким образом клетки, стабильно экспрессирующие крысиное анти-бычье PD-L1 антитело 6C11-3A11, переносили в среду Opti-CHO без Mtx. Путем культивирования этих клеток при встряхивании в течение 14 дней (125 об/мин, 37°C, 5% CO2) может быть получен супернатант культуры, содержащий представляющее интерес антитело. Для использования в различных экспериментах, антитело в супернатанте может быть очищено известными способами, такими как аффинная хроматография или ионообменная хроматография.
[Пример 3]
1. Введение
С целью создания нового способа диагностики опухолей в этом Примере получали химерное крыса-человек анти-PD-L1 антитело путем культивирования клеток яичников китайского хомяка (клетки CHO), которые экспрессируют ген химерного антитела, в котором ген вариабельной области крысиного анти-бычьего PD-L1 антитела 6С11-3A11 объединяли с геном константной области иммуноглобулина человека (IgG4).
2. Материалы и методы
2.1 Получение вектора, экспрессирующего химерное крыса-человек анти-PD-L1 антитело (Фиг.10)
Ниже показано, что химерное крыса-человека анти-PD-L1 антитело получали с использованием крысиного анти-бычьего PD-L1 моноклонального антитела 6C11-3A11 в качестве его вариабельной области.
Кратко, из гибридомы, продуцирующей крысиное анти-бычье PD-L1 антитело, 6C11-3A11, определяли гены тяжелой цепи и легкой цепи. Затем, получали нуклеотидную последовательность путем соединения генов вариабельной области тяжелой цепи и легкой цепи вышеуказанного крысиного антитела с константной областью тяжелой цепи IgG4 и константой областью легкой цепи каппа известного антитела человека, соответственно. После оптимизации кодонов проводили синтез генов так, чтобы сайт распознавания фермента рестрикции NotI, последовательность Козака, последовательность легкой цепи химерного антитела, последовательность поли-A дополнительного сигнала (PABGH), последовательность промотора (PCMV), сайт распознавания фермента рестрикции SacI, последовательность интрона (INRBG), последовательность Козака, последовательность тяжелой цепи химерного антитела и сайт распознавания фермента рестрикции XbaI были расположены в указанном порядке. Цепь синтезированного гена встраивали в вектор экспрессии pDC6 (любезно представлен профессором S. Suzuki, Research Center for Zoonosis Control, Hokkaido University) в сайт клонирования (сайты распознавания ферментов рестрикции NotI и XbaI расположены ниже PCMV и между INRBG и PABGH), используя сайты распознавания ферментов рестрикции, так что указанные выше последовательности расположены в указанном выше порядке (Фиг.10). Таким образом, контруировали вектор, экспрессирующий химерное крыса-человек анти-PD-L1антитело. Этот вектор экспрессии трансфицировали в клетки CHO-DG44 (CHO-DG44(dfhr-/-)), которые являются клетками с дефицитом дигидрофолатредуктазы, и клоны с высокой экспрессией отбирали dot-блоттингом. Для повышения экспрессии амплификацию генов можно выполнять путем добавления нагрузки на клетки в среде, содержащей 60 нМ, 250 нМ или 1000 нМ метотрексата (Mtx). Полученные таким образом клетки, стабильно экспрессирующие химерное крыса-человек анти-PD-L1 антитело 6C11-3A11, переносили в среду Opti-CHO без Mtx. Путем культивирования этих клеток при встряхивании в течение 14 дней (125 об/мин, 37°C, 5% CO2) может быть получен супернатант культуры, содержащий представляющее интерес антитело. Для использования в различных экспериментах, антитело в супернатанте может быть очищено известными способами, такими как аффинная хроматография или ионообменная хроматография.
[Пример 4]
1. Введение
Что касается PD-L1 в опухолях собак, ранее был описан способ детекции иммуногистохимическим окрашиванием крысиного анти-бычьего анти-PD-L1 антитела 6G7-E1, и профили экспрессии в различных опухолях (Maekawa N, Konnai S, Okagawa T, Ikebuchi R , Izumi Y, Takagi S, Kagawa Y, Nakajima C, Suzuki Y, Kato Y, Murata S, Ohashi K. PLoS One. 2016 Jun 11(6): e0157176). В рассматриваемом примере, для оценки повышенной эффективности крысиного анти-бычьего PD-L1 антитела 6C11-3A11 по сравнению с существующии анти-PD-L1 антителом 6G7-E1 в анализе экспрессии PD-L1 в опухолях собак, осуществляли иммуногистохимические окрашивания различных опухолей собак для непосредственного сравнение чувствительности детекции PD-L1 6G7-E1 и 6C11-3A11.
2. Материалы и методы
2.1 Сравнение методом проточной цитометрии при использовании клеток, стабильно экспрессирующих PD-L1-EGFP собаки, CHO-DG44 (Фиг.11)
Во-первых, для получения клеток, экспрессирующих мембранные PD-L1 собак, в клетки CHO-DG44 в количестве 4×106 вводили 2,5 мкг плазмиды экспрессии PD-L1-EGFP (pEGFP-N2-cPD-L1), полученной в разделе 2,3 Примера 1 или pEGFP-N2 (отрицательный контроль), используя липофектамин LTX (Invitrogen). Через сорок восемь часов среду меняли на среду CD DG44 (Life Technologies), содержащую G418 (Enzo Life Science) в количестве 800 мкг/мл, добавку GlutaMAX (Life Technologies) в количестве 20 мл/л и 10% Pluronic F-68 (Life Technologies) в количестве 18 мл/л, а затем отбирали стабильно экспрессирующие клетки и клонировали путем ограниченого разведения. Полученную таким образом клетку, экспрессирующую мембранные PD-L1 собак, или клетку, экспрессирующую EGFP, подвергали взаимодействию с крысиным анти-бычьим PD-L1 антителом 6C11-3A11 или 6G7-E1 при комнатной температуре в течение 30 мин. После промывки осуществляли дектецию связывания антител на клеточной поверхности с помощью козлиного анти-крысиного Ig антитела, меченного аллофикоцианином (Beckman Coulter). Для анализа использовали FACS Verse (Becton, Dickinson and Company). В качестве отрицательного контрольного использовали IgG2a (κ) или изотипический контроль IgM (κ) (BD Bioscience). Для каждой операции промывки и разведения антител использовали 10% инактивированную козью сыворотку с добавлением PBS.
Результаты приведены на Фиг.11. Крысиные анти-бычьи анти-PD-L1 антитела 6C11-3A11 и 6G7-E1 специфически связывались с клетками, экспрессирующими мембранные PD-L1 собак. Полученная интенсивность флуоресценции была выше для 6C11-3A11, чем с 6G7-E1, что указывает на то, что 6C11-3A11 является антителом с более высоким уровнем сродства.
2.2 Сравнение чувствительности обнаружения обоих антител в анализе экспрессии PD-L1 различных опухолей собак (иммуногистохимическое окрашивание)
Согласно методике, описанной в разделе 2.6 Примера 1 осуществляли окрашивание крысиным анти-бычьим антителом PD-L1 6C11-3A11, используя образцы плоскоклеточного рака кожи собак (n=5), аденокарциномы носы (n=5), переходноклеточной карциномы (n=5), рака анального мешка (n=5), саркомы мягких тканей (n=5) и остеосаркомы (n=5). Иммуногистохимическое окрашивание, используя крысиное анти-бычье антитело PD-L1 6G7-E1 проводили аналогично, используя срезы тех же аналитов. Конечная концентрация 6G7-E1, используемая в этом случае, составила 10 мкг/мл, и в качестве второго антитела использовали меченное биотином козье антитело против крысы (Jackson ImmunoResearch Laboratories).
Результаты приведены на Фиг.12 и 13. Несмотря на то, что специфические сигналы не наблюдали при плоскоклеточной карциномы, аденокарциноме носа, переходно-клеточной карциноме и раке анального мешка и саркоме мягких тканей при окрашивании 6G7-E1, удовлетворительные положительные реакции получали при окрашивании 6C11-3A11. С другой стороны, специфические сигналы также были получены с 6G7-E1 при остеосаркоме, но окрашивание 6C11-3A11 давало более высокую интенсивность сигнала. Положительный прогностический показатель по PD-L1 этих опухолей, полученных при окрашивании 6C11-3A11, составила 100% (5 из 5 случаев) во всех вышеперечисленных видах опухолей, за исключением саркомы мягких тканей, которая имела положительный прогностический показатель PD-L1, равный 80% (4 из 5 случаев).
Затем, иммуногистохимически окрашивали образцы злокачественной меланомы полости рта (n=17), аденокарциномы молочной железы (n=10), гистиоцитарной саркомы (n=10), диффузной крупноклеточной лимфомы (n=10) и трансмиссивной венерической опухоли (n=4) с помщью 6C11-3A11 для анализа экспрессии в PD-L1.
Результаты показаны на Фиг.14. Положительный прогностический показатель по PD-L1 составила 100% (17 из 17 случаев), 100% при аденокарциноме молочной железы (10 из 10 случаев), 20% при гистиоцитарной саркоме (2 из 10 случаев), 20% при диффузной крупноклеточной В-клеточной лимфоме (2 из 10 случаев) и 0% при трансмиссивной венерической опухоли (0 из 4 случаев).
Вышеописанные результаты показали, что 6C11-3A11 превосходит существующее анти-PD-L1 антитело 6G7-E1 для детекции PD-L1 собаки.
[Пример 5]
1. Введение
Болезнь Джона является хронической инфекцией крупного рогатого скота, вызванная Mycobacterium avium subsp. paratuberculosis. У крупного рогатого скота, пораженного болезнью Джона, экспрессия PD-L1 была подтверждена в клетках, инфицированных M. avium subsp. paratuberculosis, в поражениях подвздошной кишки, которые расположены в месте инфицирования этой бактерией (Okagawa T, Konnai S, Nishimori A, Ikebuchi R, Mizorogi S, Nagata R, Kawaji S, Tanaka S, Kagawa Y, Murata S, Mori Y and Ohashi K. Infect Immun, 84:77-89, 2016). В рассматриваемом примере иммуногистохимическое окрашивание поражений подвздошной кишки крупного рогатого скота с болезнью Джона проводили для проверки того может ли крысиное анти-бычье антитело PD-L1 6C11-3A11 использоваться для детекции бычьего PD-L1 или нет.
2. Материалы и методы
2.1. Конструирование клеток, экспрессирующих PD-L1 крупного рогатого скота
Нуклеотидная последовательность полноразмерной кДНК гена PD-L1 крупного рогатого скота (GenBank accession number AB510902; Ikebuchi R, Konnai S, Shirai T, Sunden Y, Murata S, Onuma M, Ohashi K. Vet. Res. 2011 Sep. 26; 42:103). На основе информации о нуклеотидной последовательности получали клетки, экспрессирующие бычий PD-L1 на мембране. Для получения плазмиды экспрессии бычьего PD-L1, осуществляли ПЦР, используя в качестве матрицы синтезированную кДНК из PBMC быка и праймеры, сконструированные путем добавления сайтов распознавания NheI и XhoI на 5'-конец (boPD-L1-EGFP F и R). Продукты ПЦР расщепляли с помощью NheI (Takara) и XhoI (Takara), очищали с помощью набора для гель экстракции Fastgene Gel/PCR Extraction (NIPPON Genetics) и клонировали в вектор pEGFP-N2 (Clontech), который подвергали аналогичной обработке ферментами рестрикции. Полученную интересующую плазмиду экспрессии экстрагировали, используя набор QIAGEN Plasmid Midi kit (Qiagen), и хранили при -30°C перед использованием в экспериментах. Далее, полученная таким образом плазмида экспрессии обозначена как pEGFP-N2-boPD-L1.
Праймер (boPD-L1-EGFP F): CTAGCTAGCACCATGAGGATATATAGTGTCTTAAC (SEQ ID NO:124)
Праймер (boPD-L1-EGFP R): CAATCTCGAGTTACAGACAGAAGATGACTGC (SEQ ID NO:125)
Клетки, экспрессирующие бычий PD-L1 на мембране, получали методом, описанным ниже. Сначала, 2,5 мкг pEGFP-N2-boPD-L1 или pEGFP-N2 (отрицательный контроль) вводили в клетки 4×106 CHO-DG44, используя Lipofectamine LTX (Invitrogen). Через сорок восемь часов среду меняли на среду CD DG44 (Life Technologies), содержащую G418 (Enzo Life Science) в количестве 800 мкг/мл, добавку GlutaMAX (Life Technologies) в количестве 20 мл/л и 10% Pluronic F-68 (Life Technologies) в количестве 18 мл/л; затем, осуществляли селекцию одновременной с клонированием путем ограниченного разведения (клетки, эксперессирующие бычьи PD-L1, и клетки, экспрессирующие EGFP). Для подтверждения экспрессии бычьих PD-L1 в полученных таким образом клетках, экспрессирующих бычьи PD-L1, визуализировали внутриклеточную локализацию EGFP с помощью инфертированного конфокального лазерного микроскопа LSM700 (ZEISS).
2.2. Специфичность связывания крысиного анти-бычьего PD-L1 антитела 6C11-3A11 с бычьим PD-L1
С помощью проточной цитометрии было подтверждено, что крысиное анти-бычье антитело PD-L1 6C11-3A11 специфически связывается с клеткой, экспрессирующей бычий PD-L1 (описано выше). Во-первых, крысиное анти-бычье PD-L1 антитело 6C11-3A11 или изотипический контроль крысиного IgG2a (κ) (BD Biosciences) в качестве негативного контроля взаимодействовал с клетками, эксперссирующими бычьи PD-L1, или клетками, эксперссирующими EGFP (негативный контроль) при комнатной температуре в течение 30 мин. После промывки APC-меченое анти-крысиное IgG антитело козы (Southern Biotech) взаимодействовало при комнатной температуре в течение 30 минут. После промывки антитела, связанного с клеточной поверхностью, детектировали с помощью FACS Verse (BD Biosciences). Для каждой операции промывки и разведения антител использовали PBS, дополненный 1% бычьим сывороточным альбумином (Sigma-Aldrich).
Результаты показаны на Фиг.15. Было обнаружено, что крысиное анти-бычье антитело PD-L1 6C11-3A11 специфически связывается с клеткой, экспрессирующей бычий PD-L1.
2.3. Иммуногистохимическое окрашивание c использованием образцов тканей инфицированного крупного рогатого скота
Для подтверждения того, что крысиное анти-бычье антитело PD-L1 6C11-3A11 можно использовать для иммуногистохимического окрашивания по PD-L1 тканей крупного рогатого скота, иммуногистохимическое окрашивание проводили с помощью фиксированных формалином образцов бычьей ткани в парафине. Вкратце, для окрашивания использовали фиксированную подвздошную ткань крупного рогатого скота с естественным образом приобретенной болезнью Джона (№ 1, представляющий клинические симптомы болезни Джона, такие как диарея и сильное истощение), крупного рогатого скота, экспериментально зараженного M. avium subsp. paratuberculosis (№ 65, клинические симптомы, такие как распространение M. avium subsp. paratuberculosis и диарея); Okagawa T, Konnai S, Nishimori A, Ikebuchi R, Mizorogi S, Nagata R, Kawaji S, Tanaka S, Kagawa Y, Murata S, Mori Y and Ohashi K. Infect Immun, 84:77-89, 2016) и инфицированного контрольного крупно рогатого скота (C№6) (фиксаты любезно представлены Dr. Yasuyuki Mori, National Institute of Animal Health, National Agriculture and Food Research Organization). Согласно общепринятым методам, окрашенные образцы депарафинизировали и затем подвергали микроволновой обработке (5 мин, дважды) в цитратном буфере. Затем проводили реакцию образцов с крысиным анти-бычьим антителом PD-L1 6C11-3A11 (400-кратное разведение) в течение 30 мин, а затем с Simple Stain Mouse MAX-PO (Rat) (Nichirei Bioscience) в течение 30 мин. Наконец, проводили реакцию образцов с диаминобензидином (DAB) в течение 10 мин для окрашивания, а затем наблюдали под оптическим микроскопом.
Результаты приведены на Фиг.16. Крысиное анти-бычье антитело PD-L1 6C11-3A11 детектировало экспрессию PD-L1 в клетках, инфицированных M. avium subsp. paratuberculosis (подтверждено окрашиванием Циля-Нильсена) в очагах поражения подвздошной кишки крупного рогатого скота № 1 при естественном развитии болезни Джона и у крупного рогатого скота с эксперементально инфицированной болезнью, № 65 (Фиг. 16а, b). С другой стороны, PD-L1 не экспрессировался в подвздошной кишке неинфицированного крупного рогатого скота (C№6), поэтому реакция крысиного анти-бычьего антитела PD-L1 6C11-3A11 (неспецифическая реакция) не распознавалась (Фиг.16а).
Как описано выше, было показано, что крысиное анти-бычье антитело PD-L1 6C11-3A11 может быть использовано для детекции PD-L1 в тканях крупного рогатого скота при иммуногистохимическом окрашивании.
Все публикации, патенты и патентные заявки, цитируемые в настоящем документе, включены в него посредством ссылки в полном объеме.
Промышенная применимость
Анти-PD-L1 антитело по настоящему изобретению можно использоваться для диагностики злокачественных новообразований и/или инфекций. Кроме того, анти-PD-L1 антитело по настоящему изобретения также можно использовать для селекции животных, подходящих для терапии антителами против PD-L1.
Перечень последовательностей
<SEQ ID NO:1>
SEQ ID NO:1 соответствует аминокислотной последовательности CDR1 вариабельной области легкой цепи (VL) анти-PD-L1 антитела 6C11-3A11 (IgG2a).
KSISKY
<SEQ ID NO:2>
SEQ ID NO:2 соответствует аминокислотной последовательности CDR3 of the VL анти-PD-L1 антитела 6C11-3A11 (IgG2a).
QQHNEYPLT
<SEQ ID NO:3>
SEQ ID NO:3 соответствует аминокислотной последовательности CDR1 вариабельной области тяжелой цепи (VH) анти-PD-L1 антитела 6C11-3A11 (IgG2a).
GYTFTDYI
<SEQ ID NO:4>
SEQ ID NO:4 соответствует аминокислотной последовательности CDR2 VH анти-PD-L1 антитела 6C11-3A11 (IgG2a).
INPDSGGN
<SEQ ID NO:5>
SEQ ID NO:5 соответствует аминокислотной последовательности CDR3 VH анти-PD-L1 антитела 6C11-3A11 (IgG2a).
ARGITMMVVISHWKFDF
<SEQ ID NO:6>
SEQ ID NO:6 соответствует аминокислотной последовательности the VL анти-PD-L1 антитела 6C11-3A11 (IgG2a).
MRVQIQFWGLLLLWTSGIQCDVQMTQSPSNLAASPGESVSINCKASKSISKYLAWYQQKPGKANKLLIYSGSTLQSGTPSRFSGSGSGTDFTLTIRNLEPEDFGLYYCQQHNEYPLTFGSGTKLEIK
<SEQ ID NO:7>
SEQ ID NO:7 соответствует аминокислотной последовательности VH анти-PD-L1 антитела 6C11-3A11 (IgG2a).
MGWICIIFLVAIATGAHSQVKLLQSGAALVKPGDSVKMSCKASGYTFTDYIIHWVKQSHGKSLEWIGYINPDSGGNNYNEKFKSKATLTVDKSSSTAYMEFSRLTSEDSAIYYCARGITMMVVISHWKFDFWGPGTMVTVSS
<SEQ ID NO:8>
SEQ ID NO:8 соответствует аминокислотной последовательности константной области легкой цепи (каппа) анти-PD-L1 антитела 6C11-3A11 (IgG2a).
RADAAPTVSIFPPSTEQLATGGASVVCLMNNFYPRDISVKWKIDGTERRDGVLDSVTDQDSKDSTYSMSSTLSLTKADYESHNLYTCEVVHKTSSSPVVKSFNRNEC*
<SEQ ID NO:9>
SEQ ID NO:9 соответствует аминокислотной последовательности константной области тяжелой цепи (CH) анти-PD-L1 антитела 6C11-3A11 (IgG2a).
AETTAPSVYPLAPGTALKSNSMVTLGCLVKGYFPEPVTVTWNSGALSSGVHTFPAVLQSGLYTLTSSVTVPSSTWSSQAVTCNVAHPASSTKVDKKIVPRECNPCGCTGSEVSSVFIFPPKTKDVLTITLTPKVTCVVVDISQNDPEVRFSWFIDDVEVHTAQTHAPEKQSNSTLRSVSELPIVHRDWLNGKTFKCKVNSGAFPAPIEKSISKPEGTPRGPQVYTMAPPKEEMTQSQVSITCMVKGFYPPDIYTEWKMNGQPQENYKNTPPTMDTDGSYFLYSKLNVKKETWQQGNTFTCSVLHEGLHNHHTEKSLSHSPGK*
<SEQ ID NO:10>
SEQ ID NO:10 соответствует аминокислотной последовательности (GenBank: #V01241.1) констатной области легкой цепи (каппа) антитела крысы (IgG2a).
ADAAPTVSIFPPSTEQLATGGASVVCLMNNFYPRDISVKWKIDGTERRDGVLDSVTDQDSKDSTYSMSSTLSLTKADYESHNLYTCEVVHKTSSSPVVKSFNRNEC*
<SEQ ID NO:11>
SEQ ID NO:11 соответствует аминокислотной последовательности (GenBank: #X16129.1) констатной области легкой цепи (каппа) антитела крысы (IgG2a).
RADAAPTVSIFPPSTEQLATGGASVVCLMNNFYPRDISVKWKIDGTERRDGVLDSVTDQDSKDSTYSMSSTLSLSKADYESHNLYTCEVVHKTSSSPVVKSFNRNEC
<SEQ ID NO:12>
SEQ ID NO:12 соответствует аминокислотной последовательности (GenBank: #DQ402471.1) констатной области легкой цепи (каппа) антитела крысы (IgG2a).
AAPTVSIFPPSMEQLTSGGATVVCFVNNFYPRDISVKWKIDGSEQRDGVLDSVTDQDSKDSTYSMSSTLSLTKVEYERHNLYTCEVVHKTSSSPVVKSFNRNEC*
<SEQ ID NO:13>
SEQ ID NO:13 соответствует аминокислотной последовательности (GenBank: #DQ402472.1) CH антителеа крысы (IgG2a).
APSVYPLAPGTALKSNSMVTLGCLVKGYFPEPVTVTWNSGALSSGVHTFPAVLQSGLYTLTSSVTVPSSTWSSQAVTCNVAHPASSTKVDKKIVPRECNPCGCTGSEVSSVFIFPPKTKDVLTITLTPKVTCVVVDISQNDPEVRFSWFIDDVEVHTAQTHAPEKQSNSTLRSVSELPIVHRDWLNGKTFKCKVNSGAFPAPIEKSISKPEGTPRGPQVYTMAPPKEEMTQSQVSITCMVKGFYPPDIYTEWKMNGQPQENYKNTPPTMDTDGSYFLYSKLNVKKETWQQGNTFTCSVLHEGLHNHHTEKSLSHSPGK*
<SEQ ID NO:14>
SEQ ID NO:14 соответствует нуклеотидной последовательности VLанти-PD-L1 антитела 6C11-3A11 (IgG2a).
ATGAGGGTCCAGATTCAGTTTTGGGGGCTTCTTCTGCTCTGGACATCAGGTATACAGTGTGATGTCCAGATGACCCAGTCTCCATCTAATCTTGCTGCCTCTCCTGGAGAAAGTGTTTCCATCAATTGCAAGGCAAGTAAGAGCATTAGCAAGTATTTAGCCTGGTATCAACAGAAACCTGGGAAAGCAAATAAGCTTCTTATCTACTCTGGGTCAACTTTGCAATCTGGAACTCCATCGAGGTTCAGTGGCAGTGGATCTGGTACAGATTTCACTCTCACCATCAGAAACCTGGAGCCTGAAGATTTTGGACTCTATTACTGTCAACAGCATAATGAATACCCGCTCACGTTCGGTTCTGGGACCAAGCTGGAGATCAAA
<SEQ ID NO:15>
SEQ ID NO:15 соответствует нуклеотидной последовательности VH анти-PD-L1 антитела 6C11-3A11 (IgG2a).
ATGGGATGGATCTGTATCATCTTTCTTGTGGCAATAGCTACAGGTGCCCACTCCCAGGTCAAGCTGCTGCAGTCTGGGGCTGCACTGGTGAAGCCTGGGGACTCTGTGAAGATGTCTTGCAAAGCTTCTGGTTATACATTCACTGACTACATTATACACTGGGTGAAGCAGAGTCATGGAAAAAGCCTTGAGTGGATTGGTTATATTAATCCTGACAGTGGTGGTAATAACTACAATGAAAAGTTCAAGAGCAAGGCCACATTGACTGTAGACAAATCCAGCAGCACAGCCTATATGGAGTTTAGCAGATTGACATCTGAGGATTCTGCAATCTACTACTGTGCAAGAGGGATTACCATGATGGTAGTTATTAGCCACTGGAAGTTTGACTTCTGGGGCCCAGGAACCATGGTCACCGTGTCCTCA
<SEQ ID NO:16>
SEQ ID NO:16 соответствует нуклеотидной последовательности константной области легкой цепи (каппа) анти-PD-L1 антитела 6C11-3A11 (IgG2a).
CGGGCTGATGCTGCACCAACTGTATCTATCTTCCCACCATCCACGGAACAGTTAGCAACTGGAGGTGCCTCAGTCGTGTGCCTCATGAACAACTTCTATCCCAGAGACATCAGTGTCAAGTGGAAGATTGATGGCACTGAACGACGAGATGGTGTCCTGGACAGTGTTACTGATCAGGACAGCAAAGACAGCACGTACAGCATGAGCAGCACCCTCTCGTTGACCAAGGCTGACTATGAAAGTCATAACCTCTATACCTGTGAGGTTGTTCATAAGACATCATCCTCACCCGTCGTCAAGAGCTTCAACAGGAATGAGTGTTAG
<SEQ ID NO:17>
SEQ ID NO:17 соответствует нуклеотидной последовательности CH анти-PD-L1 антитела 6C11-3A11 (IgG2a).
GCTGAAACAACAGCCCCATCTGTCTATCCACTGGCTCCTGGAACTGCTCTCAAAAGTAACTCCATGGTGACCCTGGGATGCCTGGTCAAGGGCTATTTCCCTGAGCCAGTCACCGTGACCTGGAACTCTGGAGCCCTGTCCAGCGGTGTGCACACCTTCCCAGCTGTCCTGCAGTCTGGACTCTACACTCTCACCAGCTCAGTGACTGTACCCTCCAGCACCTGGTCCAGCCAGGCCGTCACCTGCAACGTAGCCCACCCGGCCAGCAGCACCAAGGTGGACAAGAAAATTGTGCCAAGGGAATGCAATCCTTGTGGATGTACAGGCTCAGAAGTATCATCTGTCTTCATCTTCCCCCCAAAGACCAAAGATGTGCTCACCATCACTCTGACTCCTAAGGTCACGTGTGTTGTGGTAGACATTAGCCAGAATGATCCCGAGGTCCGGTTCAGCTGGTTTATAGATGACGTGGAAGTCCACACAGCTCAGACTCATGCCCCGGAGAAGCAGTCCAACAGCACTTTACGCTCAGTCAGTGAACTCCCCATCGTGCACCGGGACTGGCTCAATGGCAAGACGTTCAAATGCAAAGTCAACAGTGGAGCATTCCCTGCCCCCATCGAGAAAAGCATCTCCAAACCCGAAGGCACACCACGAGGTCCACAGGTATACACCATGGCGCCTCCCAAGGAAGAGATGACCCAGAGTCAAGTCAGTATCACCTGCATGGTAAAAGGCTTCTATCCCCCAGACATTTATACGGAGTGGAAGATGAACGGGCAGCCACAGGAAAACTACAAGAACACTCCACCTACGATGGACACAGATGGGAGTTACTTCCTCTACAGCAAGCTCAATGTAAAGAAAGAAACATGGCAGCAGGGAAACACTTTCACGTGTTCTGTGCTGCATGAGGGCCTGCACAACCACCATACTGAGAAGAGTCTCTCCCACTCTCCTGGTAAATGA
<SEQ ID NO:18>
SEQ ID NO:18 соответствует нуклеотидной последовательности (GenBank: #V01241.1) констатной области легкой цепи (каппа) антитела крысы (IgG2a).
GGGCTGATGCTGCACCAACTGTATCTATCTTCCCACCATCCACGGAACAGTTAGCAACTGGAGGTGCCTCAGTCGTGTGCCTCATGAACAACTTCTATCCCAGAGACATCAGTGTCAAGTGGAAGATTGATGGCACTGAACGACGAGATGGTGTCCTGGACAGTGTTACTGATCAGGACAGCAAAGACAGCACGTACAGCATGAGCAGCACCCTCTCGTTGACCAAGGCTGACTATGAAAGTCATAACCTCTATACCTGTGAGGTTGTTCATAAGACATCATCCTCACCCGTCGTCAAGAGCTTCAACAGGAATGAGTGTTAG
<SEQ ID NO:19>
SEQ ID NO:19 соответствует нуклеотидной последовательности (GenBank: #X16129.1) констатной области легкой цепи (каппа) антитела крысы (IgG2a).
CGGGCTGATGCTGCACCAACTGTATCTATCTTCCCACCATCCACGGAACAGTTAGCAACTGGAGGTGCCTCAGTCGTGTGCCTCATGAACAACTTCTATCCCAGAGACATCAGTGTCAAGTGGAAGATTGATGGCACTGAACGACGAGATGGTGTCCTGGACAGTGTTACTGATCAGGACAGCAAAGACAGCACGTACAGCATGAGCAGCACCCTCTCGTTGTCCAAGGCTGACTATGAAAGTCATAACCTCTATACCTGTGAGGTTGTTCATAAGACATCATCCTCACCCGTCGTCAAGAGCTTCAACAGGAATGAGTGTTAG
<SEQ ID NO:20>
SEQ ID NO:20 соответствует нуклеотидной последовательности (GenBank: #DQ402471.1) констатной области легкой цепи (каппа) антитела крысы (IgG2a).
GCCGCACCAACTGTATCCATCTTCCCACCATCCATGGAACAGTTAACATCTGGAGGTGCCACAGTCGTGTGCTTCGTGAACAACTTCTATCCCAGAGACATCAGTGTCAAGTGGAAGATTGATGGCAGTGAACAACGAGATGGTGTCCTGGACAGTGTTACTGATCAGGACAGCAAAGACAGCACGTACAGCATGAGCAGCACCCTCTCGTTGACCAAGGTTGAATATGAAAGGCATAACCTCTATACCTGTGAGGTTGTTCATAAGACATCATCCTCACCCGTCGTCAAGAGCTTCAACAGGAATGAGTGTTAG
<SEQ ID NO:21>
SEQ ID NO:21 соответствует нуклеотидной последовательности (GenBank: #DQ402472.1) CH антитела крысы (IgG2a).
CAGCCCCCTCTGTCTATCCACTGGCTCCTGGAACTGCTCTCAAAAGTAACTCCATGGTGACCCTGGGATGCCTGGTCAAGGGCTATTTCCCTGAGCCAGTCACCGTGACCTGGAACTCTGGAGCCCTGTCCAGCGGTGTGCACACCTTCCCAGCTGTCCTGCAGTCTGGACTCTACACTCTCACCAGCTCAGTGACTGTACCCTCCAGCACCTGGTCCAGCCAGGCCGTCACCTGCAACGTAGCCCACCCGGCCAGCAGCACCAAGGTGGACAAGAAAATTGTGCCAAGGGAATGCAATCCTTGTGGATGTACAGGCTCAGAAGTATCATCTGTCTTCATCTTCCCCCCAAAGACCAAAGATGTGCTCACCATCACTCTGACTCCTAAGGTCACGTGTGTTGTGGTAGACATTAGCCAGAATGATCCCGAGGTCCGGTTCAGCTGGTTTATAGATGACGTGGAAGTCCACACAGCTCAGACTCATGCCCCGGAGAAGCAGTCCAACAGCACTTTACGCTCAGTCAGTGAACTCCCCATCGTGCACCGGGACTGGCTCAATGGCAAGACGTTCAAATGCAAAGTCAACAGTGGAGCATTCCCTGCCCCCATCGAGAAAAGCATCTCCAAACCCGAAGGCACACCACGAGGTCCACAGGTATACACCATGGCGCCTCCCAAGGAAGAGATGACCCAGAGTCAAGTCAGTATCACCTGCATGGTAAAAGGCTTCTATCCCCCAGACATTTATACGGAGTGGAAGATGAACGGGCAGCCACAGGAAAACTACAAGAACACTCCACCTACGATGGACACAGATGGGAGTTACTTCCTCTACAGCAAGCTCAATGTAAAGAAAGAAACATGGCAGCAGGGAAACACTTTCACGTGTTCTGTGCTGCATGAGGGCCTGCACAACCACCATACTGAGAAGAGTCTCTCCCACTCTCCTGGTAAATGA
<SEQ ID NO:22-27>
SEQ ID NO:22-27 соответствуют нуклеотидным последовательностям праймеров cPD-L1 внутренний F, cPD-L1 внутренний R, cPD-L1 5’GSP, cPD-L1 3’GSP, cPD-L1-EGFP F and cPD-L1-EGFP R, в указанной последовательности.
<SEQ ID NO:28>
SEQ ID NO:28 соответствует аминокислотной последовательности константной области легкой цепи (каппа) антитела человека.
<SEQ ID NO:29>
SEQ ID NO:29 соответствует нуклеотидной последовательности константной области легкой цепи (каппа) антитела человека.
<SEQ ID NO:30>
SEQ ID NO:30 соответствует аминокислотной последовательности CH (CH1-CH3) антитела человека (вариант 1 IgG4).
<SEQ ID NO:31>
SEQ ID NO:31 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела человека (вариант 1 IgG4).
<SEQ ID NO:32>
SEQ ID NO:32 соответствует аминокислотной последовательности CH (CH1-CH3) антитела человека (вариант 2 IgG4).
<SEQ ID NO:33>
SEQ ID NO:33 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела человека (вариант 2 IgG4).
<SEQ ID NO:34>
SEQ ID NO:34 соответствует аминокислотной последовательности CH (CH1-CH3) антитела человека (вариант 3 IgG4).
<SEQ ID NO:35>
SEQ ID NO:35 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела человека (вариант 3 IgG4).
<SEQ ID NO:36>
SEQ ID NO:36 соответствует аминокислотной последовательности константной области легкой цепи (каппа) антитела мыши.
<SEQ ID NO:37>
SEQ ID NO:37 соответствует нуклеотидной последовательности константной области легкой цепи (каппа) антитела мыши.
<SEQ ID NO:38>
SEQ ID NO:38 соответствует аминокислотной последовательности константной области легкой цепи (каппа) антитела мыши.
<SEQ ID NO:39>
SEQ ID NO:39 соответствует нуклеотидной последовательности константной области легкой цепи (каппа) антитела мыши.
<SEQ ID NO:40>
SEQ ID NO:40 соответствует аминокислотной последовательности константной области легкой цепи (каппа) антитела мыши.
<SEQ ID NO:41>
SEQ ID NO:41 соответствует нуклеотидной последовательности константной области легкой цепи (каппа) антитела мыши.
<SEQ ID NO:42>
SEQ ID NO:42 соответствует аминокислотной последовательности константной области легкой цепи (каппа) антитела мыши.
<SEQ ID NO:43>
SEQ ID NO:43 соответствует нуклеотидной последовательности константной области легкой цепи (каппа) антитела мыши.
<SEQ ID NO:44>
SEQ ID NO:44 соответствует аминокислотной последовательности CH (CH1-CH3) антитела мыши (вариант 1 IgG1).
<SEQ ID NO:45>
SEQ ID NO:45 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела мыши (вариант 1 IgG1).
<SEQ ID NO:46>
SEQ ID NO:46 соответствует аминокислотной последовательности CH (CH1-CH3) антитела мыши (вариант 2 IgG1).
<SEQ ID NO:47>
SEQ ID NO:47 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела мыши (вариант 2 IgG1).
<SEQ ID NO:48>
SEQ ID NO:48 соответствует аминокислотной последовательности CH (CH1-CH3) антитела мыши (вариант 1 IgG2a).
<SEQ ID NO:49>
SEQ ID NO:49 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела мыши (вариант 1 IgG2a).
<SEQ ID NO:50>
SEQ ID NO:50 соответствует аминокислотной последовательности CH (CH1-CH3) антитела мыши (вариант 2 IgG2a).
<SEQ ID NO:51>
SEQ ID NO:51 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела мыши (вариант 2 IgG2a).
<SEQ ID NO:52>
SEQ ID NO:52 соответствует аминокислотной последовательности CH (CH1-CH3) антитела мыши (вариант 1 IgG2b).
<SEQ ID NO:53>
SEQ ID NO:53 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела мыши (вариант 1 IgG2b).
<SEQ ID NO:54>
SEQ ID NO:54 соответствует аминокислотной последовательности CH (CH1-CH3) антитела мыши (вариант 2 IgG2b).
<SEQ ID NO:55>
SEQ ID NO:55 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела мыши (вариант 2 IgG2b).
<SEQ ID NO:56>
SEQ ID NO:56 соответствует аминокислотной последовательности CH (CH1-CH3) антитела мыши (вариант 1 IgG2c).
<SEQ ID NO:57>
SEQ ID NO:57 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела мыши (вариант 1 IgG2c).
<SEQ ID NO:58>
SEQ ID NO:58 соответствует аминокислотной последовательности CH (CH1-CH3) антитела мыши (вариант 2 IgG2c).
<SEQ ID NO:59>
SEQ ID NO:59 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела мыши (вариант 2 IgG2c).
<SEQ ID NO:60>
SEQ ID NO:60 соответствует аминокислотной последовательности CH (CH1-CH3) антитела мыши (вариант 3 IgG2c).
<SEQ ID NO:61>
SEQ ID NO:61 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела мыши (вариант 3 IgG2c).
<SEQ ID NO:62>
SEQ ID NO:62 соответствует аминокислотной последовательности CH (CH1-CH3) антитела мыши (IgG3).
<SEQ ID NO:63>
SEQ ID NO:63 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела мыши (IgG3).
<SEQ ID NO:64>
SEQ ID NO:64 соответствует аминокислотной последовательности константной области легкой цепи (лямбда) бычьего антитела.
<SEQ ID NO:65>
SEQ ID NO:65 соответствует нуклеотидной последовательности константной области легкой цепи (лямба) бычьего антитела.
<SEQ ID NO:66>
SEQ ID NO:66 соответствует аминокислотной последовательности CH (CH1-CH3) бычьего антитела (вариант 1 IgG1).
<SEQ ID NO:67>
SEQ ID NO:67 соответствует нуклеотидной последовательности CH (CH1-CH3) бычьего антитела (вариант 1 IgG1).
<SEQ ID NO:68>
SEQ ID NO:68 соответствует аминокислотной последовательности CH (CH1-CH3) бычьего антитела (вариант 2 IgG1).
<SEQ ID NO:69>
SEQ ID NO:69 соответствует нуклеотидной последовательности CH (CH1-CH3) бычьего антитела (вариант 2 IgG1).
<SEQ ID NO:70>
SEQ ID NO:70 соответствует аминокислотной последовательности CH (CH1-CH3) бычьего антитела (вариант 3 IgG1).
<SEQ ID NO:71>
SEQ ID NO:71 соответствует нуклеотидной последовательности CH (CH1-CH3) бычьего антитела (вариант 3 IgG1).
<SEQ ID NO:72>
SEQ ID NO:72 соответствует аминокислотной последовательности CH (CH1-CH3) бычьего антитела (вариант 1 IgG2).
<SEQ ID NO:73>
SEQ ID NO:73 соответствует нуклеотидной последовательности CH (CH1-CH3) бычьего антитела (вариант 1 IgG2).
<SEQ ID NO:74>
SEQ ID NO:74 соответствует аминокислотной последовательности CH (CH1-CH3) бычьего антитела (вариант 2 IgG2).
<SEQ ID NO:75>
SEQ ID NO:75 соответствует нуклеотидной последовательности CH (CH1-CH3) бычьего антитела (вариант 2 IgG2).
<SEQ ID NO:76>
SEQ ID NO:76 соответствует аминокислотной последовательности CH (CH1-CH3) бычьего антитела (вариант 3 IgG2).
<SEQ ID NO:77>
SEQ ID NO:77 соответствует нуклеотидной последовательности CH (CH1-CH3) бычьего антитела (вариант 3 IgG2).
<SEQ ID NO:78>
SEQ ID NO:78 соответствует аминокислотной последовательности CH (CH1-CH3) бычьего антитела (вариант 1 IgG3).
<SEQ ID NO:79>
SEQ ID NO:79 соответствует нуклеотидной последовательности CH (CH1-CH3) бычьего антитела (вариант 1 IgG3).
<SEQ ID NO:80>
SEQ ID NO:80 соответствует аминокислотной последовательности CH (CH1-CH3) бычьего антитела (вариант 2 IgG3).
<SEQ ID NO:81>
SEQ ID NO:81 соответствует нуклеотидной последовательности CH (CH1-CH3) бычьего антитела (вариант 2 IgG3).
<SEQ ID NO:82>
SEQ ID NO:82 соответствует аминокислотной последовательности константной области легкой цепи (лямбда) антитела собаки.
<SEQ ID NO:83>
SEQ ID NO:83 соответствует нуклеотидной последовательности константной области легкой цепи (лямба) антитела собаки.
<SEQ ID NO:84>
SEQ ID NO:84 соответствует аминокислотной последовательности CH (CH1-CH3) антитела собаки (IgG-D).
<SEQ ID NO:85>
SEQ ID NO:85 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела собаки (IgG-D).
<SEQ ID NO:86>
SEQ ID NO:86 соответствует аминокислотной последовательности константной области легкой цепи (каппа) овечьего антитела.
<SEQ ID NO:87>
SEQ ID NO:87 соответствует нуклеотидной последовательности константной области легкой цепи (каппа) овечьего антитела.
<SEQ ID NO:88>
SEQ ID NO:88 соответствует аминокислотной последовательности константной области легкой цепи (лямбда) овечьего антитела.
<SEQ ID NO:89>
SEQ ID NO:89 соответствует нуклеотидной последовательности константной области легкой цепи (лямба) овечьего антитела.
<SEQ ID NO:90>
SEQ ID NO:90 соответствует аминокислотной последовательности CH (CH1-CH3) овечьего антитела (IgG1).
<SEQ ID NO:91>
SEQ ID NO:91 соответствует нуклеотидной последовательности CH (CH1-CH3) овечьего антитела (IgG1).
<SEQ ID NO:92>
SEQ ID NO:92 соответствует аминокислотной последовательности CH (CH1-CH3) овечьего антитела (IgG2).
<SEQ ID NO:93>
SEQ ID NO:93 соответствует нуклеотидной последовательности CH (CH1-CH3) овечьего антитела (IgG2).
<SEQ ID NO:94>
SEQ ID NO:94 соответствует аминокислотной последовательности CH (CH1-CH3) антитела свиньи (IgG1a).
<SEQ ID NO:95>
SEQ ID NO:95 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела свиньи (IgG1a).
<SEQ ID NO:96>
SEQ ID NO:96 соответствует аминокислотной последовательности CH (CH1-CH3) антитела свиньи (IgG1b).
<SEQ ID NO:97>
SEQ ID NO:97 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела свиньи (IgG1b).
<SEQ ID NO:98>
SEQ ID NO:98 соответствует аминокислотной последовательности CH (CH1-CH3) антитела свиньи (IgG2a).
<SEQ ID NO:99>
SEQ ID NO:99 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела свиньи (IgG2a).
<SEQ ID NO:100>
SEQ ID NO:100 соответствует аминокислотной последовательности CH (CH1-CH3) антитела свиньи (IgG2b).
<SEQ ID NO:101>
SEQ ID NO:101 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела свиньи (IgG2b).
<SEQ ID NO:102>
SEQ ID NO:102 соответствует аминокислотной последовательности CH (CH1-CH3) антитела свиньи (IgG3).
<SEQ ID NO:103>
SEQ ID NO:103 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела свиньи (IgG3).
<SEQ ID NO:104>
SEQ ID NO:104 соответствует аминокислотной последовательности CH (CH1-CH3) антитела свиньи (IgG4a).
<SEQ ID NO:105>
SEQ ID NO:105 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела свиньи (IgG4a).
<SEQ ID NO:106>
SEQ ID NO:106 соответствует аминокислотной последовательности CH (CH1-CH3) антитела свиньи (IgG4b).
<SEQ ID NO:107>
SEQ ID NO:107 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела свиньи (IgG4b).
<SEQ ID NO:108>
SEQ ID NO:108 соответствует аминокислотной последовательности CH (CH1-CH3) антитела свиньи (IgG5a).
<SEQ ID NO:109>
SEQ ID NO:109 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела свиньи (IgG5a).
<SEQ ID NO:110>
SEQ ID NO:110 соответствует аминокислотной последовательности CH (CH1-CH3) антитела свиньи (IgG5b).
<SEQ ID NO:111>
SEQ ID NO:111 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела свиньи (IgG5b).
<SEQ ID NO:112>
SEQ ID NO:112 соответствует аминокислотной последовательности CH (CH1-CH3) антитела свиньи (IgG6a).
<SEQ ID NO:113>
SEQ ID NO:113 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела свиньи (IgG6a).
<SEQ ID NO:114>
SEQ ID NO:114 соответствует аминокислотной последовательности CH (CH1-CH3) антитела свиньи (IgG6b).
<SEQ ID NO:115>
SEQ ID NO:115 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела свиньи (IgG6b).
<SEQ ID NO:116>
SEQ ID NO:116 соответствует аминокислотной последовательности константной области легкой цепи (CL)(определена как Ig лямбда) антитела буйвола.
<SEQ ID NO:117>
SEQ ID NO:117 соответствует нуклеотидной последовательности константной области легкой цепи (определена как Ig лямбда) (CL) антитела буйвола.
<SEQ ID NO:118>
SEQ ID NO:118 соответствует аминокислотной последовательности CH (CH1-CH3) антитела буйвола (определена как IgG1).
<SEQ ID NO:119>
SEQ ID NO:119 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела буйвола (определена как IgG1).
<SEQ ID NO:120>
SEQ ID NO:120 соответствует аминокислотной последовательности CH (CH1-CH3) антитела буйвола (определена как IgG2).
<SEQ ID NO:121>
SEQ ID NO:121 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела буйвола (определена как IgG2).
<SEQ ID NO:122>
SEQ ID NO:122 соответствует аминокислотной последовательности CH (CH1-CH3) антитела буйвола (определена как IgG3).
<SEQ ID NO:123>
SEQ ID NO:123 соответствует нуклеотидной последовательности CH (CH1-CH3) антитела буйвола (определена как IgG3).
<SEQ ID NO:124>
SEQ ID NO:124 соответствует нуклеотидной последовательности праймера boPD-L1-EGFP F.
<SEQ ID NO:125>
SEQ ID NO:125 соответствует нуклеотидной последовательности праймера boPD-L1-EGFP R.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> НЭШНЛ ЮНИВЕРСИТИ КОРПОРЕЙШН ХОККАЙДО ЮНИВЕРСИТИ
ФУСО ФАРМАСЬЮТИКАЛ ИНДАСТРИЗ, ЛТД.
<120> АНТИ-PD-L1 АНТИТЕЛО ДЛЯ ДЕТЕКЦИИ PD-L1
<130> FP-233PCT
<150> JP 2017-61389
<151> 2017-03-27
<160> 125
<170> PatentIn version 3.5
<210> 1
<211> 6
<212> Белок
<213> Bubalus arnee
<400> 1
Lys Ser Ile Ser Lys Tyr
1 5
<210> 2
<211> 9
<212> Белок
<213> Bubalus arnee
<400> 2
Gln Gln His Asn Glu Tyr Pro Leu Thr
1 5
<210> 3
<211> 8
<212> Белок
<213> Bubalus arnee
<400> 3
Gly Tyr Thr Phe Thr Asp Tyr Ile
1 5
<210> 4
<211> 8
<212> Белок
<213> Bubalus arnee
<400> 4
Ile Asn Pro Asp Ser Gly Gly Asn
1 5
<210> 5
<211> 17
<212> Белок
<213> Bubalus arnee
<400> 5
Ala Arg Gly Ile Thr Met Met Val Val Ile Ser His Trp Lys Phe Asp
1 5 10 15
Phe
<210> 6
<211> 127
<212> Белок
<213> Bubalus arnee
<400> 6
Met Arg Val Gln Ile Gln Phe Trp Gly Leu Leu Leu Leu Trp Thr Ser
1 5 10 15
Gly Ile Gln Cys Asp Val Gln Met Thr Gln Ser Pro Ser Asn Leu Ala
20 25 30
Ala Ser Pro Gly Glu Ser Val Ser Ile Asn Cys Lys Ala Ser Lys Ser
35 40 45
Ile Ser Lys Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Asn
50 55 60
Lys Leu Leu Ile Tyr Ser Gly Ser Thr Leu Gln Ser Gly Thr Pro Ser
65 70 75 80
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Arg
85 90 95
Asn Leu Glu Pro Glu Asp Phe Gly Leu Tyr Tyr Cys Gln Gln His Asn
100 105 110
Glu Tyr Pro Leu Thr Phe Gly Ser Gly Thr Lys Leu Glu Ile Lys
115 120 125
<210> 7
<211> 142
<212> Белок
<213> Bubalus arnee
<400> 7
Met Gly Trp Ile Cys Ile Ile Phe Leu Val Ala Ile Ala Thr Gly Ala
1 5 10 15
His Ser Gln Val Lys Leu Leu Gln Ser Gly Ala Ala Leu Val Lys Pro
20 25 30
Gly Asp Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr
35 40 45
Asp Tyr Ile Ile His Trp Val Lys Gln Ser His Gly Lys Ser Leu Glu
50 55 60
Trp Ile Gly Tyr Ile Asn Pro Asp Ser Gly Gly Asn Asn Tyr Asn Glu
65 70 75 80
Lys Phe Lys Ser Lys Ala Thr Leu Thr Val Asp Lys Ser Ser Ser Thr
85 90 95
Ala Tyr Met Glu Phe Ser Arg Leu Thr Ser Glu Asp Ser Ala Ile Tyr
100 105 110
Tyr Cys Ala Arg Gly Ile Thr Met Met Val Val Ile Ser His Trp Lys
115 120 125
Phe Asp Phe Trp Gly Pro Gly Thr Met Val Thr Val Ser Ser
130 135 140
<210> 8
<211> 107
<212> Белок
<213> Bubalus arnee
<400> 8
Arg Ala Asp Ala Ala Pro Thr Val Ser Ile Phe Pro Pro Ser Thr Glu
1 5 10 15
Gln Leu Ala Thr Gly Gly Ala Ser Val Val Cys Leu Met Asn Asn Phe
20 25 30
Tyr Pro Arg Asp Ile Ser Val Lys Trp Lys Ile Asp Gly Thr Glu Arg
35 40 45
Arg Asp Gly Val Leu Asp Ser Val Thr Asp Gln Asp Ser Lys Asp Ser
50 55 60
Thr Tyr Ser Met Ser Ser Thr Leu Ser Leu Thr Lys Ala Asp Tyr Glu
65 70 75 80
Ser His Asn Leu Tyr Thr Cys Glu Val Val His Lys Thr Ser Ser Ser
85 90 95
Pro Val Val Lys Ser Phe Asn Arg Asn Glu Cys
100 105
<210> 9
<211> 322
<212> Белок
<213> Bubalus arnee
<400> 9
Ala Glu Thr Thr Ala Pro Ser Val Tyr Pro Leu Ala Pro Gly Thr Ala
1 5 10 15
Leu Lys Ser Asn Ser Met Val Thr Leu Gly Cys Leu Val Lys Gly Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Thr Trp Asn Ser Gly Ala Leu Ser Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Gly Leu Tyr Thr Leu
50 55 60
Thr Ser Ser Val Thr Val Pro Ser Ser Thr Trp Ser Ser Gln Ala Val
65 70 75 80
Thr Cys Asn Val Ala His Pro Ala Ser Ser Thr Lys Val Asp Lys Lys
85 90 95
Ile Val Pro Arg Glu Cys Asn Pro Cys Gly Cys Thr Gly Ser Glu Val
100 105 110
Ser Ser Val Phe Ile Phe Pro Pro Lys Thr Lys Asp Val Leu Thr Ile
115 120 125
Thr Leu Thr Pro Lys Val Thr Cys Val Val Val Asp Ile Ser Gln Asn
130 135 140
Asp Pro Glu Val Arg Phe Ser Trp Phe Ile Asp Asp Val Glu Val His
145 150 155 160
Thr Ala Gln Thr His Ala Pro Glu Lys Gln Ser Asn Ser Thr Leu Arg
165 170 175
Ser Val Ser Glu Leu Pro Ile Val His Arg Asp Trp Leu Asn Gly Lys
180 185 190
Thr Phe Lys Cys Lys Val Asn Ser Gly Ala Phe Pro Ala Pro Ile Glu
195 200 205
Lys Ser Ile Ser Lys Pro Glu Gly Thr Pro Arg Gly Pro Gln Val Tyr
210 215 220
Thr Met Ala Pro Pro Lys Glu Glu Met Thr Gln Ser Gln Val Ser Ile
225 230 235 240
Thr Cys Met Val Lys Gly Phe Tyr Pro Pro Asp Ile Tyr Thr Glu Trp
245 250 255
Lys Met Asn Gly Gln Pro Gln Glu Asn Tyr Lys Asn Thr Pro Pro Thr
260 265 270
Met Asp Thr Asp Gly Ser Tyr Phe Leu Tyr Ser Lys Leu Asn Val Lys
275 280 285
Lys Glu Thr Trp Gln Gln Gly Asn Thr Phe Thr Cys Ser Val Leu His
290 295 300
Glu Gly Leu His Asn His His Thr Glu Lys Ser Leu Ser His Ser Pro
305 310 315 320
Gly Lys
<210> 10
<211> 106
<212> Белок
<213> Bubalus arnee
<400> 10
Ala Asp Ala Ala Pro Thr Val Ser Ile Phe Pro Pro Ser Thr Glu Gln
1 5 10 15
Leu Ala Thr Gly Gly Ala Ser Val Val Cys Leu Met Asn Asn Phe Tyr
20 25 30
Pro Arg Asp Ile Ser Val Lys Trp Lys Ile Asp Gly Thr Glu Arg Arg
35 40 45
Asp Gly Val Leu Asp Ser Val Thr Asp Gln Asp Ser Lys Asp Ser Thr
50 55 60
Tyr Ser Met Ser Ser Thr Leu Ser Leu Thr Lys Ala Asp Tyr Glu Ser
65 70 75 80
His Asn Leu Tyr Thr Cys Glu Val Val His Lys Thr Ser Ser Ser Pro
85 90 95
Val Val Lys Ser Phe Asn Arg Asn Glu Cys
100 105
<210> 11
<211> 107
<212> Белок
<213> Bubalus arnee
<400> 11
Arg Ala Asp Ala Ala Pro Thr Val Ser Ile Phe Pro Pro Ser Thr Glu
1 5 10 15
Gln Leu Ala Thr Gly Gly Ala Ser Val Val Cys Leu Met Asn Asn Phe
20 25 30
Tyr Pro Arg Asp Ile Ser Val Lys Trp Lys Ile Asp Gly Thr Glu Arg
35 40 45
Arg Asp Gly Val Leu Asp Ser Val Thr Asp Gln Asp Ser Lys Asp Ser
50 55 60
Thr Tyr Ser Met Ser Ser Thr Leu Ser Leu Ser Lys Ala Asp Tyr Glu
65 70 75 80
Ser His Asn Leu Tyr Thr Cys Glu Val Val His Lys Thr Ser Ser Ser
85 90 95
Pro Val Val Lys Ser Phe Asn Arg Asn Glu Cys
100 105
<210> 12
<211> 104
<212> Белок
<213> Bubalus arnee
<400> 12
Ala Ala Pro Thr Val Ser Ile Phe Pro Pro Ser Met Glu Gln Leu Thr
1 5 10 15
Ser Gly Gly Ala Thr Val Val Cys Phe Val Asn Asn Phe Tyr Pro Arg
20 25 30
Asp Ile Ser Val Lys Trp Lys Ile Asp Gly Ser Glu Gln Arg Asp Gly
35 40 45
Val Leu Asp Ser Val Thr Asp Gln Asp Ser Lys Asp Ser Thr Tyr Ser
50 55 60
Met Ser Ser Thr Leu Ser Leu Thr Lys Val Glu Tyr Glu Arg His Asn
65 70 75 80
Leu Tyr Thr Cys Glu Val Val His Lys Thr Ser Ser Ser Pro Val Val
85 90 95
Lys Ser Phe Asn Arg Asn Glu Cys
100
<210> 13
<211> 318
<212> Белок
<213> Bubalus arnee
<400> 13
Ala Pro Ser Val Tyr Pro Leu Ala Pro Gly Thr Ala Leu Lys Ser Asn
1 5 10 15
Ser Met Val Thr Leu Gly Cys Leu Val Lys Gly Tyr Phe Pro Glu Pro
20 25 30
Val Thr Val Thr Trp Asn Ser Gly Ala Leu Ser Ser Gly Val His Thr
35 40 45
Phe Pro Ala Val Leu Gln Ser Gly Leu Tyr Thr Leu Thr Ser Ser Val
50 55 60
Thr Val Pro Ser Ser Thr Trp Ser Ser Gln Ala Val Thr Cys Asn Val
65 70 75 80
Ala His Pro Ala Ser Ser Thr Lys Val Asp Lys Lys Ile Val Pro Arg
85 90 95
Glu Cys Asn Pro Cys Gly Cys Thr Gly Ser Glu Val Ser Ser Val Phe
100 105 110
Ile Phe Pro Pro Lys Thr Lys Asp Val Leu Thr Ile Thr Leu Thr Pro
115 120 125
Lys Val Thr Cys Val Val Val Asp Ile Ser Gln Asn Asp Pro Glu Val
130 135 140
Arg Phe Ser Trp Phe Ile Asp Asp Val Glu Val His Thr Ala Gln Thr
145 150 155 160
His Ala Pro Glu Lys Gln Ser Asn Ser Thr Leu Arg Ser Val Ser Glu
165 170 175
Leu Pro Ile Val His Arg Asp Trp Leu Asn Gly Lys Thr Phe Lys Cys
180 185 190
Lys Val Asn Ser Gly Ala Phe Pro Ala Pro Ile Glu Lys Ser Ile Ser
195 200 205
Lys Pro Glu Gly Thr Pro Arg Gly Pro Gln Val Tyr Thr Met Ala Pro
210 215 220
Pro Lys Glu Glu Met Thr Gln Ser Gln Val Ser Ile Thr Cys Met Val
225 230 235 240
Lys Gly Phe Tyr Pro Pro Asp Ile Tyr Thr Glu Trp Lys Met Asn Gly
245 250 255
Gln Pro Gln Glu Asn Tyr Lys Asn Thr Pro Pro Thr Met Asp Thr Asp
260 265 270
Gly Ser Tyr Phe Leu Tyr Ser Lys Leu Asn Val Lys Lys Glu Thr Trp
275 280 285
Gln Gln Gly Asn Thr Phe Thr Cys Ser Val Leu His Glu Gly Leu His
290 295 300
Asn His His Thr Glu Lys Ser Leu Ser His Ser Pro Gly Lys
305 310 315
<210> 14
<211> 381
<212> ДНК
<213> Bubalus arnee
<400> 14
atgagggtcc agattcagtt ttgggggctt cttctgctct ggacatcagg tatacagtgt 60
gatgtccaga tgacccagtc tccatctaat cttgctgcct ctcctggaga aagtgtttcc 120
atcaattgca aggcaagtaa gagcattagc aagtatttag cctggtatca acagaaacct 180
gggaaagcaa ataagcttct tatctactct gggtcaactt tgcaatctgg aactccatcg 240
aggttcagtg gcagtggatc tggtacagat ttcactctca ccatcagaaa cctggagcct 300
gaagattttg gactctatta ctgtcaacag cataatgaat acccgctcac gttcggttct 360
gggaccaagc tggagatcaa a 381
<210> 15
<211> 426
<212> ДНК
<213> Bubalus arnee
<400> 15
atgggatgga tctgtatcat ctttcttgtg gcaatagcta caggtgccca ctcccaggtc 60
aagctgctgc agtctggggc tgcactggtg aagcctgggg actctgtgaa gatgtcttgc 120
aaagcttctg gttatacatt cactgactac attatacact gggtgaagca gagtcatgga 180
aaaagccttg agtggattgg ttatattaat cctgacagtg gtggtaataa ctacaatgaa 240
aagttcaaga gcaaggccac attgactgta gacaaatcca gcagcacagc ctatatggag 300
tttagcagat tgacatctga ggattctgca atctactact gtgcaagagg gattaccatg 360
atggtagtta ttagccactg gaagtttgac ttctggggcc caggaaccat ggtcaccgtg 420
tcctca 426
<210> 16
<211> 324
<212> ДНК
<213> Bubalus arnee
<400> 16
cgggctgatg ctgcaccaac tgtatctatc ttcccaccat ccacggaaca gttagcaact 60
ggaggtgcct cagtcgtgtg cctcatgaac aacttctatc ccagagacat cagtgtcaag 120
tggaagattg atggcactga acgacgagat ggtgtcctgg acagtgttac tgatcaggac 180
agcaaagaca gcacgtacag catgagcagc accctctcgt tgaccaaggc tgactatgaa 240
agtcataacc tctatacctg tgaggttgtt cataagacat catcctcacc cgtcgtcaag 300
agcttcaaca ggaatgagtg ttag 324
<210> 17
<211> 969
<212> ДНК
<213> Bubalus arnee
<400> 17
gctgaaacaa cagccccatc tgtctatcca ctggctcctg gaactgctct caaaagtaac 60
tccatggtga ccctgggatg cctggtcaag ggctatttcc ctgagccagt caccgtgacc 120
tggaactctg gagccctgtc cagcggtgtg cacaccttcc cagctgtcct gcagtctgga 180
ctctacactc tcaccagctc agtgactgta ccctccagca cctggtccag ccaggccgtc 240
acctgcaacg tagcccaccc ggccagcagc accaaggtgg acaagaaaat tgtgccaagg 300
gaatgcaatc cttgtggatg tacaggctca gaagtatcat ctgtcttcat cttcccccca 360
aagaccaaag atgtgctcac catcactctg actcctaagg tcacgtgtgt tgtggtagac 420
attagccaga atgatcccga ggtccggttc agctggttta tagatgacgt ggaagtccac 480
acagctcaga ctcatgcccc ggagaagcag tccaacagca ctttacgctc agtcagtgaa 540
ctccccatcg tgcaccggga ctggctcaat ggcaagacgt tcaaatgcaa agtcaacagt 600
ggagcattcc ctgcccccat cgagaaaagc atctccaaac ccgaaggcac accacgaggt 660
ccacaggtat acaccatggc gcctcccaag gaagagatga cccagagtca agtcagtatc 720
acctgcatgg taaaaggctt ctatccccca gacatttata cggagtggaa gatgaacggg 780
cagccacagg aaaactacaa gaacactcca cctacgatgg acacagatgg gagttacttc 840
ctctacagca agctcaatgt aaagaaagaa acatggcagc agggaaacac tttcacgtgt 900
tctgtgctgc atgagggcct gcacaaccac catactgaga agagtctctc ccactctcct 960
ggtaaatga 969
<210> 18
<211> 323
<212> ДНК
<213> Bubalus arnee
<400> 18
gggctgatgc tgcaccaact gtatctatct tcccaccatc cacggaacag ttagcaactg 60
gaggtgcctc agtcgtgtgc ctcatgaaca acttctatcc cagagacatc agtgtcaagt 120
ggaagattga tggcactgaa cgacgagatg gtgtcctgga cagtgttact gatcaggaca 180
gcaaagacag cacgtacagc atgagcagca ccctctcgtt gaccaaggct gactatgaaa 240
gtcataacct ctatacctgt gaggttgttc ataagacatc atcctcaccc gtcgtcaaga 300
gcttcaacag gaatgagtgt tag 323
<210> 19
<211> 324
<212> ДНК
<213> Bubalus arnee
<400> 19
cgggctgatg ctgcaccaac tgtatctatc ttcccaccat ccacggaaca gttagcaact 60
ggaggtgcct cagtcgtgtg cctcatgaac aacttctatc ccagagacat cagtgtcaag 120
tggaagattg atggcactga acgacgagat ggtgtcctgg acagtgttac tgatcaggac 180
agcaaagaca gcacgtacag catgagcagc accctctcgt tgtccaaggc tgactatgaa 240
agtcataacc tctatacctg tgaggttgtt cataagacat catcctcacc cgtcgtcaag 300
agcttcaaca ggaatgagtg ttag 324
<210> 20
<211> 315
<212> ДНК
<213> Bubalus arnee
<400> 20
gccgcaccaa ctgtatccat cttcccacca tccatggaac agttaacatc tggaggtgcc 60
acagtcgtgt gcttcgtgaa caacttctat cccagagaca tcagtgtcaa gtggaagatt 120
gatggcagtg aacaacgaga tggtgtcctg gacagtgtta ctgatcagga cagcaaagac 180
agcacgtaca gcatgagcag caccctctcg ttgaccaagg ttgaatatga aaggcataac 240
ctctatacct gtgaggttgt tcataagaca tcatcctcac ccgtcgtcaa gagcttcaac 300
aggaatgagt gttag 315
<210> 21
<211> 959
<212> ДНК
<213> Bubalus arnee
<400> 21
cagccccctc tgtctatcca ctggctcctg gaactgctct caaaagtaac tccatggtga 60
ccctgggatg cctggtcaag ggctatttcc ctgagccagt caccgtgacc tggaactctg 120
gagccctgtc cagcggtgtg cacaccttcc cagctgtcct gcagtctgga ctctacactc 180
tcaccagctc agtgactgta ccctccagca cctggtccag ccaggccgtc acctgcaacg 240
tagcccaccc ggccagcagc accaaggtgg acaagaaaat tgtgccaagg gaatgcaatc 300
cttgtggatg tacaggctca gaagtatcat ctgtcttcat cttcccccca aagaccaaag 360
atgtgctcac catcactctg actcctaagg tcacgtgtgt tgtggtagac attagccaga 420
atgatcccga ggtccggttc agctggttta tagatgacgt ggaagtccac acagctcaga 480
ctcatgcccc ggagaagcag tccaacagca ctttacgctc agtcagtgaa ctccccatcg 540
tgcaccggga ctggctcaat ggcaagacgt tcaaatgcaa agtcaacagt ggagcattcc 600
ctgcccccat cgagaaaagc atctccaaac ccgaaggcac accacgaggt ccacaggtat 660
acaccatggc gcctcccaag gaagagatga cccagagtca agtcagtatc acctgcatgg 720
taaaaggctt ctatccccca gacatttata cggagtggaa gatgaacggg cagccacagg 780
aaaactacaa gaacactcca cctacgatgg acacagatgg gagttacttc ctctacagca 840
agctcaatgt aaagaaagaa acatggcagc agggaaacac tttcacgtgt tctgtgctgc 900
atgagggcct gcacaaccac catactgaga agagtctctc ccactctcct ggtaaatga 959
<210> 22
<211> 20
<212> ДНК
<213> Bubalus arnee
<400> 22
atgagaatgt ttagtgtctt 20
<210> 23
<211> 24
<212> ДНК
<213> Bubalus arnee
<400> 23
ttatgtctct tcaaattgta tatc 24
<210> 24
<211> 17
<212> ДНК
<213> Bubalus arnee
<400> 24
ttttagacag aaagtga 17
<210> 25
<211> 20
<212> ДНК
<213> Bubalus arnee
<400> 25
gaccagctct tcttggggaa 20
<210> 26
<211> 26
<212> ДНК
<213> Bubalus arnee
<400> 26
gaagatctat gagaatgttt agtgtc 26
<210> 27
<211> 28
<212> ДНК
<213> Bubalus arnee
<400> 27
ggaattctgt ctcttcaaat tgtatatc 28
<210> 28
<211> 106
<212> Белок
<213> Bubalus arnee
<400> 28
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
1 5 10 15
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
20 25 30
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
35 40 45
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
50 55 60
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
65 70 75 80
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
85 90 95
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
100 105
<210> 29
<211> 321
<212> ДНК
<213> Bubalus arnee
<400> 29
actgtggctg caccatctgt cttcatcttc ccgccatctg atgagcagtt gaaatctgga 60
actgcctctg ttgtgtgcct gctgaataac ttctatccca gagaggccaa agtacagtgg 120
aaggtggata acgccctcca atcgggtaac tcccaggaga gtgtcacaga gcaggacagc 180
aaggacagca cctacagcct cagcagcacc ctgacgctga gcaaagcaga ctacgagaaa 240
cacaaagtct acgcctgcga agtcacccat cagggcctga gctcgcccgt cacaaagagc 300
ttcaacaggg gagagtgtta g 321
<210> 30
<211> 229
<212> Белок
<213> Bubalus arnee
<400> 30
Glu Ser Lys Tyr Gly Pro Pro Cys Pro Ser Cys Pro Ala Pro Glu Phe
1 5 10 15
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
20 25 30
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
35 40 45
Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val
50 55 60
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser
65 70 75 80
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
85 90 95
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
100 105 110
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
115 120 125
Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln
130 135 140
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
145 150 155 160
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
165 170 175
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu
180 185 190
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
195 200 205
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
210 215 220
Leu Ser Leu Gly Lys
225
<210> 31
<211> 690
<212> ДНК
<213> Bubalus arnee
<400> 31
gagtccaaat atggtccccc atgcccatca tgcccagcac ctgagttcct ggggggacca 60
tcagtcttcc tgttcccccc aaaacccaag gacactctca tgatctcccg gacccctgag 120
gtcacgtgcg tggtggtgga cgtgagccag gaagaccccg aggtccagtt caactggtac 180
gtggatggcg tggaggtgca taatgccaag acaaagccgc gggaggagca gttcaacagc 240
acgtaccgtg tggtcagcgt cctcaccgtc ctgcaccagg actggctgaa cggcaaggag 300
tacaagtgca aggtctccaa caaaggcctc ccgtcctcca tcgagaaaac catctccaaa 360
gccaaagggc agccccgaga gccacaggtg tacaccctgc ccccatccca ggaggagatg 420
accaagaacc aggtcagcct gacctgcctg gtcaaaggct tctaccccag cgacatcgcc 480
gtggagtggg agagcaatgg gcagccggag aacaactaca agaccacgcc tcccgtgctg 540
gactccgacg gctccttctt cctctacagc aggctaaccg tggacaagag caggtggcag 600
gaggggaatg tcttctcatg ctccgtgatg catgaggctc tgcacaacca ctacacacag 660
aagagcctct ccctgtctct gggtaaatga 690
<210> 32
<211> 229
<212> Белок
<213> Bubalus arnee
<400> 32
Glu Ser Lys Tyr Gly Pro Pro Cys Pro Ser Cys Pro Ala Pro Glu Phe
1 5 10 15
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
20 25 30
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
35 40 45
Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val
50 55 60
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser
65 70 75 80
Thr Tyr Arg Val Val Ser Val Leu Thr Val Val His Gln Asp Trp Leu
85 90 95
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
100 105 110
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
115 120 125
Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln
130 135 140
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
145 150 155 160
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
165 170 175
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu
180 185 190
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
195 200 205
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
210 215 220
Leu Ser Leu Gly Lys
225
<210> 33
<211> 690
<212> ДНК
<213> Bubalus arnee
<400> 33
gagtccaaat atggtccccc gtgcccatca tgcccagcac ctgagttcct ggggggacca 60
tcagtcttcc tgttcccccc aaaacccaag gacactctca tgatctcccg gacccctgag 120
gtcacgtgcg tggtggtgga cgtgagccag gaagaccccg aggtccagtt caactggtac 180
gtggatggcg tggaggtgca taatgccaag acaaagccgc gggaggagca gttcaacagc 240
acgtaccgtg tggtcagcgt cctcaccgtc gtgcaccagg actggctgaa cggcaaggag 300
tacaagtgca aggtctccaa caaaggcctc ccgtcctcca tcgagaaaac catctccaaa 360
gccaaagggc agccccgaga gccacaggtg tacaccctgc ccccatccca ggaggagatg 420
accaagaacc aggtcagcct gacctgcctg gtcaaaggct tctaccccag cgacatcgcc 480
gtggagtggg agagcaatgg gcagccggag aacaactaca agaccacgcc tcccgtgctg 540
gactccgacg gctccttctt cctctacagc aggctaaccg tggacaagag caggtggcag 600
gaggggaatg tcttctcatg ctccgtgatg catgaggctc tgcacaacca ctacacgcag 660
aagagcctct ccctgtctct gggtaaatga 690
<210> 34
<211> 217
<212> Белок
<213> Bubalus arnee
<400> 34
Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
1 5 10 15
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
20 25 30
Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr
35 40 45
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
50 55 60
Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
65 70 75 80
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
85 90 95
Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
100 105 110
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met
115 120 125
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
130 135 140
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
145 150 155 160
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
165 170 175
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val
180 185 190
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
195 200 205
Lys Ser Leu Ser Leu Ser Leu Gly Lys
210 215
<210> 35
<211> 654
<212> ДНК
<213> Bubalus arnee
<400> 35
gcacctgagt tcctgggggg accatcagtc ttcctgttcc ccccaaaacc caaggacact 60
ctcatgatct cccggacccc tgaggtcacg tgcgtggtgg tggacgtgag ccaggaagac 120
cccgaggtcc agttcaactg gtacgtggat ggcgtggagg tgcataatgc caagacaaag 180
ccgcgggagg agcagttcaa cagcacgtac cgtgtggtca gcgtcctcac cgtcctgcac 240
caggactggc tgaacggcaa ggagtacaag tgcaaggtct ccaacaaagg cctcccgtcc 300
tccatcgaga aaaccatctc caaagccaaa gggcagcccc gagagccaca ggtgtacacc 360
ctgcccccat cccaggagga gatgaccaag aaccaggtca gcctgacctg cctggtcaaa 420
ggcttctacc ccagcgacat cgccgtggag tgggagagca atgggcagcc ggagaacaac 480
tacaagacca cgcctcccgt gctggactcc gacggctcct tcttcctcta cagcaagctc 540
accgtggaca agagcaggtg gcaggagggg aacgtcttct catgctccgt gatgcatgag 600
gctctgcaca accactacac gcagaagagc ctctccctgt ctctgggtaa atga 654
<210> 36
<211> 106
<212> Белок
<213> Bubalus arnee
<400> 36
Ala Asp Ala Ala Pro Thr Val Ser Ile Phe Pro Pro Ser Ser Glu Gln
1 5 10 15
Leu Thr Ser Gly Gly Ala Ser Val Val Cys Phe Leu Asn Asn Phe Tyr
20 25 30
Pro Lys Asp Ile Asn Val Lys Trp Lys Ile Asp Gly Ser Glu Arg Gln
35 40 45
Asn Gly Val Leu Asn Ser Trp Thr Asp Gln Asp Ser Lys Asp Ser Thr
50 55 60
Tyr Ser Met Ser Ser Thr Leu Thr Leu Thr Lys Asp Glu Tyr Glu Arg
65 70 75 80
His Asn Ser Tyr Thr Cys Glu Ala Thr His Lys Thr Ser Thr Ser Pro
85 90 95
Ile Val Lys Ser Phe Asn Arg Asn Glu Cys
100 105
<210> 37
<211> 321
<212> ДНК
<213> Bubalus arnee
<400> 37
gctgatgctg caccaactgt atccatcttc ccaccatcca gtgagcagtt aacatctgga 60
ggtgcctcag tcgtgtgctt cttgaacaac ttctacccca aagacatcaa tgtcaagtgg 120
aagattgatg gcagtgaacg acaaaatggc gtcctgaaca gttggactga tcaggacagc 180
aaagacagca cctacagcat gagcagcacc ctcacgttga ccaaggacga gtatgaacga 240
cataacagct atacctgtga ggccactcac aagacatcaa cttcacccat tgtcaagagc 300
ttcaacagga atgagtgtta g 321
<210> 38
<211> 106
<212> Белок
<213> Bubalus arnee
<400> 38
Gly Gln Pro Lys Ser Ser Pro Ser Val Thr Leu Phe Pro Pro Ser Ser
1 5 10 15
Glu Glu Leu Glu Thr Asn Lys Ala Thr Leu Val Cys Thr Ile Thr Asp
20 25 30
Phe Tyr Pro Gly Val Val Thr Val Asp Trp Lys Val Asp Gly Thr Pro
35 40 45
Val Thr Gln Gly Met Glu Thr Thr Gln Pro Ser Lys Gln Ser Asn Asn
50 55 60
Lys Tyr Met Ala Ser Ser Tyr Leu Thr Leu Thr Ala Arg Ala Trp Glu
65 70 75 80
Arg His Ser Ser Tyr Ser Cys Gln Val Thr His Glu Gly His Thr Val
85 90 95
Glu Lys Ser Leu Ser Arg Ala Asp Cys Ser
100 105
<210> 39
<211> 321
<212> ДНК
<213> Bubalus arnee
<400> 39
ggccagccca agtcttcgcc atcagtcacc ctgtttccac cttcctctga agagctcgag 60
actaacaagg ccacactggt gtgtacgatc actgatttct acccaggtgt ggtgacagtg 120
gactggaagg tagatggtac ccctgtcact cagggtatgg agacaaccca gccttccaaa 180
cagagcaaca acaagtacat ggctagcagc tacctgaccc tgacagcaag agcatgggaa 240
aggcatagca gttacagctg ccaggtcact catgaaggtc acactgtgga gaagagtttg 300
tcccgtgctg actgttccta g 321
<210> 40
<211> 105
<212> Белок
<213> Bubalus arnee
<400> 40
Gly Gln Pro Lys Ser Thr Pro Thr Leu Thr Val Phe Pro Pro Ser Ser
1 5 10 15
Glu Glu Leu Lys Glu Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asn
20 25 30
Phe Ser Pro Ser Gly Val Thr Val Ala Trp Lys Ala Asn Gly Thr Pro
35 40 45
Ile Thr Gln Gly Val Asp Thr Ser Asn Pro Thr Lys Glu Gly Asn Lys
50 55 60
Phe Met Ala Ser Ser Phe Leu His Leu Thr Ser Asp Gln Trp Arg Ser
65 70 75 80
His Asn Ser Phe Thr Cys Gln Val Thr His Glu Gly Asp Thr Val Glu
85 90 95
Lys Ser Leu Ser Pro Ala Glu Cys Leu
100 105
<210> 41
<211> 318
<212> ДНК
<213> Bubalus arnee
<400> 41
ggtcagccca agtccactcc cactctcacc gtgtttccac cttcctctga ggagctcaag 60
gaaaacaaag ccacactggt gtgtctgatt tccaactttt ccccgagtgg tgtgacagtg 120
gcctggaagg caaatggtac acctatcacc cagggtgtgg acacttcaaa tcccaccaaa 180
gagggcaaca agttcatggc cagcagcttc ctacatttga catcggacca gtggagatct 240
cacaacagtt ttacctgtca agttacacat gaaggggaca ctgtggagaa gagtctgtct 300
cctgcagaat gtctctaa 318
<210> 42
<211> 105
<212> Белок
<213> Bubalus arnee
<400> 42
Gly Gln Pro Lys Ser Thr Pro Thr Leu Thr Met Phe Pro Pro Ser Pro
1 5 10 15
Glu Glu Leu Gln Glu Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asn
20 25 30
Phe Ser Pro Ser Gly Val Thr Val Ala Trp Lys Ala Asn Gly Thr Pro
35 40 45
Ile Thr Gln Gly Val Asp Thr Ser Asn Pro Thr Lys Glu Asp Asn Lys
50 55 60
Tyr Met Ala Ser Ser Phe Leu His Leu Thr Ser Asp Gln Trp Arg Ser
65 70 75 80
His Asn Ser Phe Thr Cys Gln Val Thr His Glu Gly Asp Thr Val Glu
85 90 95
Lys Ser Leu Ser Pro Ala Glu Cys Leu
100 105
<210> 43
<211> 318
<212> ДНК
<213> Bubalus arnee
<400> 43
ggtcagccca agtccactcc cacactcacc atgtttccac cttcccctga ggagctccag 60
gaaaacaaag ccacactcgt gtgtctgatt tccaattttt ccccaagtgg tgtgacagtg 120
gcctggaagg caaatggtac acctatcacc cagggtgtgg acacttcaaa tcccaccaaa 180
gaggacaaca agtacatggc cagcagcttc ttacatttga catcggacca gtggagatct 240
cacaacagtt ttacctgcca agttacacat gaaggggaca ctgtggagaa gagtctgtct 300
cctgcagaat gtctctaa 318
<210> 44
<211> 324
<212> Белок
<213> Bubalus arnee
<400> 44
Ala Lys Thr Thr Pro Pro Ser Val Tyr Pro Leu Ala Pro Gly Ser Ala
1 5 10 15
Ala Gln Thr Asn Ser Met Val Thr Leu Gly Cys Leu Val Lys Gly Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Thr Trp Asn Ser Gly Ser Leu Ser Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Glu Ser Asp Leu Tyr Thr Leu
50 55 60
Ser Ser Ser Val Thr Val Pro Ser Ser Pro Arg Pro Ser Glu Thr Val
65 70 75 80
Thr Cys Asn Val Ala His Pro Ala Ser Ser Thr Lys Val Asp Lys Lys
85 90 95
Ile Val Pro Arg Asp Cys Gly Cys Lys Pro Cys Ile Cys Thr Val Pro
100 105 110
Glu Val Ser Ser Val Phe Ile Phe Pro Pro Lys Pro Lys Asp Val Leu
115 120 125
Thr Ile Thr Leu Thr Pro Lys Val Thr Cys Val Val Val Asp Ile Ser
130 135 140
Lys Asp Asp Pro Glu Val Gln Phe Ser Trp Phe Val Asp Asp Val Glu
145 150 155 160
Val His Thr Ala Gln Thr Gln Pro Arg Glu Glu Gln Phe Asn Ser Thr
165 170 175
Phe Arg Ser Val Ser Glu Leu Pro Ile Met His Gln Asp Trp Leu Asn
180 185 190
Gly Lys Glu Phe Lys Cys Arg Val Asn Ser Ala Ala Phe Pro Ala Pro
195 200 205
Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Arg Pro Lys Ala Pro Gln
210 215 220
Val Tyr Thr Ile Pro Pro Pro Lys Glu Gln Met Ala Lys Asp Lys Val
225 230 235 240
Ser Leu Thr Cys Met Ile Thr Asp Phe Phe Pro Glu Asp Ile Thr Val
245 250 255
Glu Trp Gln Trp Asn Gly Gln Pro Ala Glu Asn Tyr Lys Asn Thr Gln
260 265 270
Pro Ile Met Asn Thr Asn Gly Ser Tyr Phe Val Tyr Ser Lys Leu Asn
275 280 285
Val Gln Lys Ser Asn Trp Glu Ala Gly Asn Thr Phe Thr Cys Ser Val
290 295 300
Leu His Glu Gly Leu His Asn His His Thr Glu Lys Ser Leu Ser His
305 310 315 320
Ser Pro Gly Lys
<210> 45
<211> 975
<212> ДНК
<213> Bubalus arnee
<400> 45
gccaaaacga cacccccatc tgtctatcca ctggcccctg gatctgctgc ccaaactaac 60
tccatggtga ccctgggatg cctggtcaag ggctatttcc ctgagccagt gacagtgacc 120
tggaactctg gatccctgtc cagcggtgtg cacaccttcc cagctgtcct ggagtctgac 180
ctctacactc tgagcagctc agtgactgtc ccctccagcc ctcggcccag cgagaccgtc 240
acctgcaacg ttgcccaccc ggccagcagc accaaggtgg acaagaaaat tgtgcccagg 300
gattgtggtt gtaagccttg catatgtaca gtcccagaag tatcatctgt cttcatcttc 360
cccccaaagc ccaaggatgt gctcaccatt actctgactc ctaaggtcac gtgtgttgtg 420
gtagacatca gcaaggatga tcccgaggtc cagttcagct ggtttgtaga tgatgtggag 480
gtgcacacag ctcagacgca accccgggag gagcagttca acagcacttt ccgctcagtc 540
agtgaacttc ccatcatgca ccaggactgg ctcaatggca aggagttcaa atgcagggtc 600
aacagtgcag ctttccctgc ccccatcgag aaaaccatct ccaaaaccaa aggcagaccg 660
aaggctccac aggtgtacac cattccacct cccaaggagc agatggccaa ggataaagtc 720
agtctgacct gcatgataac agacttcttc cctgaagaca ttactgtgga gtggcagtgg 780
aatgggcagc cagcggagaa ctacaagaac actcagccca tcatgaacac gaatggctct 840
tacttcgtct acagcaagct caatgtgcag aagagcaact gggaggcagg aaatactttc 900
acctgctctg tgttacatga gggcctgcac aaccaccata ctgagaagag cctctcccac 960
tctcctggta aatga 975
<210> 46
<211> 324
<212> Белок
<213> Bubalus arnee
<400> 46
Ala Lys Thr Thr Pro Pro Ser Val Tyr Pro Leu Ala Pro Gly Ser Ala
1 5 10 15
Ala Gln Thr Asn Ser Met Val Thr Leu Gly Cys Leu Val Lys Gly Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Thr Trp Asn Ser Gly Ser Leu Ser Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Asp Leu Tyr Thr Leu
50 55 60
Ser Ser Ser Val Thr Val Pro Ser Ser Thr Trp Pro Ser Gln Thr Val
65 70 75 80
Thr Cys Asn Val Ala His Pro Ala Ser Ser Thr Lys Val Asp Lys Lys
85 90 95
Ile Val Pro Arg Asp Cys Gly Cys Lys Pro Cys Ile Cys Thr Val Pro
100 105 110
Glu Val Ser Ser Val Phe Ile Phe Pro Pro Lys Pro Lys Asp Val Leu
115 120 125
Thr Ile Thr Leu Thr Pro Lys Val Thr Cys Val Val Val Asp Ile Ser
130 135 140
Lys Asp Asp Pro Glu Val Gln Phe Ser Trp Phe Val Asp Asp Val Glu
145 150 155 160
Val His Thr Ala Gln Thr Lys Pro Arg Glu Glu Gln Ile Asn Ser Thr
165 170 175
Phe Arg Ser Val Ser Glu Leu Pro Ile Met His Gln Asp Trp Leu Asn
180 185 190
Gly Lys Glu Phe Lys Cys Arg Val Asn Ser Ala Ala Phe Pro Ala Pro
195 200 205
Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Arg Pro Lys Ala Pro Gln
210 215 220
Val Tyr Thr Ile Pro Pro Pro Lys Glu Gln Met Ala Lys Asp Lys Val
225 230 235 240
Ser Leu Thr Cys Met Ile Thr Asn Phe Phe Pro Glu Asp Ile Thr Val
245 250 255
Glu Trp Gln Trp Asn Gly Gln Pro Ala Glu Asn Tyr Lys Asn Thr Gln
260 265 270
Pro Ile Met Asp Thr Asp Gly Ser Tyr Phe Val Tyr Ser Lys Leu Asn
275 280 285
Val Gln Lys Ser Asn Trp Glu Ala Gly Asn Thr Phe Thr Cys Ser Val
290 295 300
Leu His Glu Gly Leu His Asn His His Thr Glu Lys Ser Leu Ser His
305 310 315 320
Ser Pro Gly Lys
<210> 47
<211> 975
<212> ДНК
<213> Bubalus arnee
<400> 47
gccaaaacga cacccccatc tgtctatcca ctggcccctg gatctgctgc ccaaactaac 60
tccatggtga ccctgggatg cctggtcaag ggctatttcc ctgagccagt gacagtgacc 120
tggaactctg gatccctgtc cagcggtgtg cacaccttcc cagctgtcct gcagtctgac 180
ctctacactc tgagcagctc agtgactgtc ccctccagca cctggcccag ccagaccgtc 240
acctgcaacg ttgcccaccc ggccagcagc accaaggtgg acaagaaaat tgtgcccagg 300
gattgtggtt gtaagccttg catatgtaca gtcccagaag tatcatctgt cttcatcttc 360
cccccaaagc ccaaggatgt gctcaccatt actctgactc ctaaggtcac gtgtgttgtg 420
gtagacatca gcaaggatga tcccgaggtc cagttcagct ggtttgtaga tgatgtggag 480
gtgcacacag ctcagacgaa accccgggag gagcagatca acagcacttt ccgttcagtc 540
agtgaacttc ccatcatgca ccaggactgg ctcaatggca aggagttcaa atgcagggtc 600
aacagtgcag ctttccctgc ccccatcgag aaaaccatct ccaaaaccaa aggcagaccg 660
aaggctccac aggtgtacac cattccacct cccaaggagc agatggccaa ggataaagtc 720
agtctgacct gcatgataac aaacttcttc cctgaagaca ttactgtgga gtggcagtgg 780
aatgggcagc cagcggagaa ctacaagaac actcagccca tcatggacac agatggctct 840
tacttcgtct acagcaagct caatgtgcag aagagcaact gggaggcagg aaatactttc 900
acctgctctg tgttacatga gggcctgcac aaccaccata ctgagaagag cctctcccac 960
tctcctggta aatga 975
<210> 48
<211> 330
<212> Белок
<213> Bubalus arnee
<400> 48
Ala Lys Thr Thr Ala Pro Ser Val Tyr Pro Leu Ala Pro Val Cys Gly
1 5 10 15
Asp Thr Thr Gly Ser Ser Val Thr Leu Gly Cys Leu Val Lys Gly Tyr
20 25 30
Phe Pro Glu Pro Val Thr Leu Thr Trp Asn Ser Gly Ser Leu Ser Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Asp Leu Tyr Thr Leu
50 55 60
Ser Ser Ser Val Thr Val Thr Ser Ser Thr Trp Pro Ser Gln Ser Ile
65 70 75 80
Thr Cys Asn Val Ala His Pro Ala Ser Ser Thr Lys Val Asp Lys Lys
85 90 95
Ile Glu Pro Arg Gly Pro Thr Ile Lys Pro Cys Pro Pro Cys Lys Cys
100 105 110
Pro Ala Pro Asn Leu Leu Gly Gly Pro Ser Val Phe Ile Phe Pro Pro
115 120 125
Lys Ile Lys Asp Val Leu Met Ile Ser Leu Ser Pro Ile Val Thr Cys
130 135 140
Val Val Val Asp Val Ser Glu Asp Asp Pro Asp Val Gln Ile Ser Trp
145 150 155 160
Phe Val Asn Asn Val Glu Val His Thr Ala Gln Thr Gln Thr His Arg
165 170 175
Glu Asp Tyr Asn Ser Thr Leu Arg Val Val Ser Ala Leu Pro Ile Gln
180 185 190
His Gln Asp Trp Met Ser Gly Lys Glu Phe Lys Cys Lys Val Asn Asn
195 200 205
Lys Asp Leu Pro Ala Pro Ile Glu Arg Thr Ile Ser Lys Pro Lys Gly
210 215 220
Ser Val Arg Ala Pro Gln Val Tyr Val Leu Pro Pro Pro Glu Glu Glu
225 230 235 240
Met Thr Lys Lys Gln Val Thr Leu Thr Cys Met Val Thr Asp Phe Met
245 250 255
Pro Glu Asp Ile Tyr Val Glu Trp Thr Asn Asn Gly Lys Thr Glu Leu
260 265 270
Asn Tyr Lys Asn Thr Glu Pro Val Leu Asp Ser Asp Gly Ser Tyr Phe
275 280 285
Met Tyr Ser Lys Leu Arg Val Glu Lys Lys Asn Trp Val Glu Arg Asn
290 295 300
Ser Tyr Ser Cys Ser Val Val His Glu Gly Leu His Asn His His Thr
305 310 315 320
Thr Lys Ser Phe Ser Arg Thr Pro Gly Lys
325 330
<210> 49
<211> 993
<212> ДНК
<213> Bubalus arnee
<400> 49
gccaaaacaa cagccccatc ggtctatcca ctggcccctg tgtgtggaga tacaactggc 60
tcctcggtga ctctaggatg cctggtcaag ggttatttcc ctgagccagt gaccttgacc 120
tggaactctg gatccctgtc cagtggtgtg cacaccttcc cagctgtcct gcagtctgac 180
ctctacaccc tcagcagctc agtgactgta acctcgagca cctggcccag ccagtccatc 240
acctgcaatg tggcccaccc ggcaagcagc accaaggtgg acaagaaaat tgagcccaga 300
gggcccacaa tcaagccctg tcctccatgc aaatgcccag cacctaacct cttgggtgga 360
ccatccgtct tcatcttccc tccaaagatc aaggatgtac tcatgatctc cctgagcccc 420
atagtcacat gtgtggtggt ggatgtgagc gaggatgacc cagatgtcca gatcagctgg 480
tttgtgaaca acgtggaagt acacacagct cagacacaaa cccatagaga ggattacaac 540
agtactctcc gggtggtcag tgccctcccc atccagcacc aggactggat gagtggcaag 600
gagttcaaat gcaaggtcaa caacaaagac ctcccagcgc ccatcgagag aaccatctca 660
aaacccaaag ggtcagtaag agctccacag gtatatgtct tgcctccacc agaagaagag 720
atgactaaga aacaggtcac tctgacctgc atggtcacag acttcatgcc tgaagacatt 780
tacgtggagt ggaccaacaa cgggaaaaca gagctaaact acaagaacac tgaaccagtc 840
ctggactctg atggttctta cttcatgtac agcaagctga gagtggaaaa gaagaactgg 900
gtggaaagaa atagctactc ctgttcagtg gtccacgagg gtctgcacaa tcaccacacg 960
actaagagct tctcccggac tccgggtaaa tga 993
<210> 50
<211> 330
<212> Белок
<213> Bubalus arnee
<400> 50
Ala Lys Thr Thr Ala Pro Ser Val Tyr Pro Leu Ala Pro Val Cys Gly
1 5 10 15
Asp Thr Thr Gly Ser Ser Val Thr Leu Gly Cys Leu Val Lys Gly Tyr
20 25 30
Phe Pro Glu Pro Val Thr Leu Thr Trp Asn Ser Gly Ser Leu Ser Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Asp Leu Tyr Thr Leu
50 55 60
Ser Ser Ser Val Thr Val Thr Ser Ser Thr Trp Pro Ser Gln Ser Ile
65 70 75 80
Thr Cys Asn Val Ala His Pro Ala Ser Ser Thr Lys Val Asp Lys Lys
85 90 95
Ile Glu Pro Arg Gly Pro Thr Ile Lys Pro Cys Pro Pro Cys Lys Cys
100 105 110
Pro Ala Pro Asn Leu Leu Gly Gly Pro Ser Val Phe Ile Phe Pro Pro
115 120 125
Lys Ile Lys Asp Val Leu Met Ile Ser Leu Ser Pro Met Val Thr Cys
130 135 140
Val Val Val Asp Val Ser Glu Asp Asp Pro Asp Val Gln Ile Ser Trp
145 150 155 160
Phe Val Asn Asn Val Glu Val Leu Thr Ala Gln Thr Gln Thr His Arg
165 170 175
Glu Asp Tyr Asn Ser Thr Leu Arg Val Val Ser Ala Leu Pro Ile Gln
180 185 190
His Gln Asp Trp Met Ser Gly Lys Glu Phe Lys Cys Lys Val Asn Asn
195 200 205
Lys Ala Leu Pro Ala Pro Ile Glu Arg Thr Ile Ser Lys Pro Lys Gly
210 215 220
Ser Val Arg Ala Pro Gln Val Tyr Val Leu Pro Pro Pro Glu Glu Glu
225 230 235 240
Met Thr Lys Lys Gln Val Thr Leu Thr Cys Met Val Thr Asp Phe Met
245 250 255
Pro Glu Asp Ile Tyr Val Glu Trp Thr Asn Asn Gly Lys Thr Glu Leu
260 265 270
Asn Tyr Lys Asn Thr Glu Pro Val Leu Asp Ser Asp Gly Ser Tyr Phe
275 280 285
Met Tyr Ser Lys Leu Arg Val Glu Lys Lys Asn Trp Val Glu Arg Asn
290 295 300
Ser Tyr Ser Cys Ser Val Val His Glu Gly Leu His Asn His His Thr
305 310 315 320
Thr Lys Ser Phe Ser Arg Thr Pro Gly Lys
325 330
<210> 51
<211> 993
<212> ДНК
<213> Bubalus arnee
<400> 51
gccaaaacaa cagccccatc ggtctatcca ctggcccctg tgtgtggaga tacaactggc 60
tcctcggtga ctctaggatg cctggtcaag ggttatttcc ctgagccagt gaccttgacc 120
tggaactctg gatccctgtc cagtggtgtg cacaccttcc cagctgtcct gcagtctgac 180
ctctacaccc tcagcagctc agtgactgta acctcgagca cctggcccag ccagtccatc 240
acctgcaatg tggcccaccc ggcaagcagc accaaggtgg acaagaaaat tgagcccaga 300
gggcccacaa tcaagccctg tcctccatgc aaatgcccag cacctaacct cttgggtgga 360
ccatccgtct tcatcttccc tccaaagatc aaggatgtac tcatgatctc cctgagtccc 420
atggtcacat gtgtggtggt ggatgtgagc gaggatgacc cagatgtcca gatcagctgg 480
ttcgtgaaca acgtggaagt actcacagct cagacacaaa cccatagaga ggattacaac 540
agtactctcc gggtggtcag tgccctcccc atccagcacc aggactggat gagtggcaag 600
gagttcaaat gcaaggtcaa caacaaagcc ctcccagcgc ccatcgagag aaccatctca 660
aaacccaaag ggtcagtaag agctccacag gtatatgtct tgcctccacc agaagaagag 720
atgactaaga aacaggtcac tctgacctgc atggtcacag acttcatgcc tgaagacatt 780
tacgtggagt ggaccaacaa cgggaaaaca gagctaaact acaagaacac tgaaccagtc 840
ctggactctg atggttctta cttcatgtac agcaagctga gagtggaaaa gaagaactgg 900
gtggaaagaa atagctactc ctgttcagtg gtccacgagg gtctgcacaa tcaccacacg 960
actaagagct tctcccggac tccgggtaaa tga 993
<210> 52
<211> 336
<212> Белок
<213> Bubalus arnee
<400> 52
Ala Lys Thr Thr Pro Pro Ser Val Tyr Pro Leu Ala Pro Gly Cys Gly
1 5 10 15
Asp Thr Thr Gly Ser Ser Val Thr Leu Gly Cys Leu Val Lys Gly Tyr
20 25 30
Phe Pro Glu Ser Val Thr Val Thr Trp Asn Ser Gly Ser Leu Ser Ser
35 40 45
Ser Val His Thr Phe Pro Ala Leu Leu Gln Ser Gly Leu Tyr Thr Met
50 55 60
Ser Ser Ser Val Thr Val Pro Ser Ser Thr Trp Pro Ser Gln Thr Val
65 70 75 80
Thr Cys Ser Val Ala His Pro Ala Ser Ser Thr Thr Val Asp Lys Lys
85 90 95
Leu Glu Pro Ser Gly Pro Ile Ser Thr Ile Asn Pro Cys Pro Pro Cys
100 105 110
Lys Glu Cys His Lys Cys Pro Ala Pro Asn Leu Glu Gly Gly Pro Ser
115 120 125
Val Phe Ile Phe Pro Pro Asn Ile Lys Asp Val Leu Met Ile Ser Leu
130 135 140
Thr Pro Lys Val Thr Cys Val Val Val Asp Val Ser Glu Asp Asp Pro
145 150 155 160
Asp Val Gln Ile Ser Trp Phe Val Asn Asn Val Glu Val His Thr Ala
165 170 175
Gln Thr Gln Thr His Arg Glu Asp Tyr Asn Ser Thr Ile Arg Val Val
180 185 190
Ser Thr Leu Pro Ile Gln His Gln Asp Trp Met Ser Gly Lys Glu Phe
195 200 205
Lys Cys Lys Val Asn Asn Lys Asp Leu Pro Ser Pro Ile Glu Arg Thr
210 215 220
Ile Ser Lys Ile Lys Gly Leu Val Arg Ala Pro Gln Val Tyr Ile Leu
225 230 235 240
Pro Pro Pro Ala Glu Gln Leu Ser Arg Lys Asp Val Ser Leu Thr Cys
245 250 255
Leu Val Val Gly Phe Asn Pro Gly Asp Ile Ser Val Glu Trp Thr Ser
260 265 270
Asn Gly His Thr Glu Glu Asn Tyr Lys Asp Thr Ala Pro Val Leu Asp
275 280 285
Ser Asp Gly Ser Tyr Phe Ile Tyr Ser Lys Leu Asn Met Lys Thr Ser
290 295 300
Lys Trp Glu Lys Thr Asp Ser Phe Ser Cys Asn Val Arg His Glu Gly
305 310 315 320
Leu Lys Asn Tyr Tyr Leu Lys Lys Thr Ile Ser Arg Ser Pro Gly Lys
325 330 335
<210> 53
<211> 1011
<212> ДНК
<213> Bubalus arnee
<400> 53
gccaaaacaa cacccccatc agtctatcca ctggcccctg ggtgtggaga tacaactggt 60
tcctccgtga ctctgggatg cctggtcaag ggctacttcc ctgagtcagt gactgtgact 120
tggaactctg gatccctgtc cagcagtgtg cacaccttcc cagctctcct gcagtctgga 180
ctctacacta tgagcagctc agtgactgtc ccctccagca cttggccaag tcagaccgtc 240
acctgcagcg ttgctcaccc agccagcagc accacggtgg acaaaaaact tgagcccagc 300
gggcccattt caacaatcaa cccctgtcct ccatgcaagg agtgtcacaa atgcccagct 360
cctaacctcg agggtggacc atccgtcttc atcttccctc caaatatcaa ggatgtactc 420
atgatctccc tgacacccaa ggtcacgtgt gtggtggtgg atgtgagcga ggatgaccca 480
gacgtccaga tcagctggtt tgtgaacaac gtggaagtac acacagctca gacacaaacc 540
catagagagg attacaacag tactatccgg gtggtcagca ccctccccat ccagcaccag 600
gactggatga gtggcaagga gttcaaatgc aaggtcaaca acaaagacct cccatcaccc 660
atcgagagaa ccatctcaaa aattaaaggg ctagtcagag ctccacaagt atacatcttg 720
ccgccaccag cagagcagtt gtccaggaaa gatgtcagtc tcacttgcct ggtcgtgggc 780
ttcaaccctg gagacatcag tgtggagtgg accagcaatg ggcatacaga ggagaactac 840
aaggacaccg caccagtcct agactctgac ggttcttact tcatatatag caagctcaat 900
atgaaaacaa gcaagtggga gaaaacagat tccttctcat gcaacgtgag acacgagggt 960
ctgaaaaatt actacctgaa gaagaccatc tcccggtctc cgggtaaatg a 1011
<210> 54
<211> 336
<212> Белок
<213> Bubalus arnee
<400> 54
Ala Lys Thr Thr Pro Pro Ser Val Tyr Pro Leu Ala Pro Gly Cys Gly
1 5 10 15
Asp Thr Thr Gly Ser Ser Val Thr Ser Gly Cys Leu Val Lys Gly Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Thr Trp Asn Ser Gly Ser Leu Ser Ser
35 40 45
Ser Val His Thr Phe Pro Ala Leu Leu Gln Ser Gly Leu Tyr Thr Met
50 55 60
Ser Ser Ser Val Thr Val Pro Ser Ser Thr Trp Pro Ser Gln Thr Val
65 70 75 80
Thr Cys Ser Val Ala His Pro Ala Ser Ser Thr Thr Val Asp Lys Lys
85 90 95
Leu Glu Pro Ser Gly Pro Ile Ser Thr Ile Asn Pro Cys Pro Pro Cys
100 105 110
Lys Glu Cys His Lys Cys Pro Ala Pro Asn Leu Glu Gly Gly Pro Ser
115 120 125
Val Phe Ile Phe Pro Pro Asn Ile Lys Asp Val Leu Met Ile Ser Leu
130 135 140
Thr Pro Lys Val Thr Cys Val Val Val Asp Val Ser Glu Asp Asp Pro
145 150 155 160
Asp Val Gln Ile Ser Trp Phe Val Asn Asn Val Glu Val His Thr Ala
165 170 175
Gln Thr Gln Thr His Arg Glu Asp Tyr Asn Ser Thr Ile Arg Val Val
180 185 190
Ser Thr Leu Pro Ile Gln His Gln Asp Trp Met Ser Gly Lys Glu Phe
195 200 205
Lys Cys Lys Val Asn Asn Lys Asp Leu Pro Ser Pro Ile Glu Arg Thr
210 215 220
Ile Ser Lys Ile Lys Gly Leu Val Arg Ala Pro Gln Val Tyr Thr Leu
225 230 235 240
Pro Pro Pro Ala Glu Gln Leu Ser Arg Lys Asp Val Ser Leu Thr Cys
245 250 255
Leu Val Val Gly Phe Asn Pro Gly Asp Ile Ser Val Glu Trp Thr Ser
260 265 270
Asn Gly His Thr Glu Glu Asn Tyr Lys Asp Thr Ala Pro Val Leu Asp
275 280 285
Ser Asp Gly Ser Tyr Phe Ile Tyr Ser Lys Leu Asn Met Lys Thr Ser
290 295 300
Lys Trp Glu Lys Thr Asp Ser Phe Ser Cys Asn Val Arg His Glu Gly
305 310 315 320
Leu Lys Asn Tyr Tyr Leu Lys Lys Thr Ile Ser Arg Ser Pro Gly Lys
325 330 335
<210> 55
<211> 1011
<212> ДНК
<213> Bubalus arnee
<400> 55
gccaaaacaa cacccccatc agtctatcca ctggcccctg ggtgtggaga tacaactggt 60
tcctccgtga cctctgggtg cctggtcaag gggtacttcc ctgagccagt gactgtgact 120
tggaactctg gatccctgtc cagcagtgtg cacaccttcc cagctctcct gcagtctgga 180
ctctacacta tgagcagctc agtgactgtc ccctccagca cctggccaag tcagaccgtc 240
acctgcagcg ttgctcaccc agccagcagc accacggtgg acaaaaaact tgagcccagc 300
gggcccattt caacaatcaa cccctgtcct ccatgcaagg agtgtcacaa atgcccagct 360
cctaacctcg agggtggacc atccgtcttc atcttccctc caaatatcaa ggatgtactc 420
atgatctccc tgacacccaa ggtcacgtgt gtggtggtgg atgtgagcga ggatgaccca 480
gacgtccaga tcagctggtt tgtgaacaac gtggaagtac acacagctca gacacaaacc 540
catagagagg attacaacag tactatccgg gtggtcagca ccctccccat ccagcaccag 600
gactggatga gtggcaagga gttcaaatgc aaggtgaaca acaaagacct cccatcaccc 660
atcgagagaa ccatctcaaa aattaaaggg ctagtcagag ctccacaagt atacactttg 720
ccgccaccag cagagcagtt gtccaggaaa gatgtcagtc tcacttgcct ggtcgtgggc 780
ttcaaccctg gagacatcag tgtggagtgg accagcaatg ggcatacaga ggagaactac 840
aaggacaccg caccagttct tgactctgac ggttcttact tcatatatag caagctcaat 900
atgaaaacaa gcaagtggga gaaaacagat tccttctcat gcaacgtgag acacgagggt 960
ctgaaaaatt actacctgaa gaagaccatc tcccggtctc cgggtaaatg a 1011
<210> 56
<211> 335
<212> Белок
<213> Bubalus arnee
<400> 56
Ala Lys Thr Thr Ala Pro Ser Val Tyr Pro Leu Ala Pro Val Cys Gly
1 5 10 15
Gly Thr Thr Gly Ser Ser Val Thr Leu Gly Cys Leu Val Lys Gly Tyr
20 25 30
Phe Pro Glu Pro Val Thr Leu Thr Trp Asn Ser Gly Ser Leu Ser Ser
35 40 45
Gly Val His Thr Phe Pro Ala Leu Leu Gln Ser Gly Leu Tyr Thr Leu
50 55 60
Ser Ser Ser Val Thr Val Thr Ser Asn Thr Trp Pro Ser Gln Thr Ile
65 70 75 80
Thr Cys Asn Val Ala His Pro Ala Ser Ser Thr Lys Val Asp Lys Lys
85 90 95
Ile Glu Pro Arg Val Pro Ile Thr Gln Asn Pro Cys Pro Pro Leu Lys
100 105 110
Glu Cys Pro Pro Cys Ala Ala Pro Asp Leu Leu Gly Gly Pro Ser Val
115 120 125
Phe Ile Phe Pro Pro Lys Ile Lys Asp Val Leu Met Ile Ser Leu Ser
130 135 140
Pro Met Val Thr Cys Val Val Val Asp Val Ser Glu Asp Asp Pro Asp
145 150 155 160
Val Gln Ile Ser Trp Phe Val Asn Asn Val Glu Val His Thr Ala Gln
165 170 175
Thr Gln Thr His Arg Glu Asp Tyr Asn Ser Thr Leu Arg Val Val Ser
180 185 190
Ala Leu Pro Ile Gln His Gln Asp Trp Met Ser Gly Lys Glu Phe Lys
195 200 205
Cys Lys Val Asn Asn Arg Ala Leu Pro Ser Pro Ile Glu Lys Thr Ile
210 215 220
Ser Lys Pro Arg Gly Pro Val Arg Ala Pro Gln Val Tyr Val Leu Pro
225 230 235 240
Pro Pro Ala Glu Glu Met Thr Lys Lys Glu Phe Ser Leu Thr Cys Met
245 250 255
Ile Thr Gly Phe Leu Pro Ala Glu Ile Ala Val Asp Trp Thr Ser Asn
260 265 270
Gly Arg Thr Glu Gln Asn Tyr Lys Asn Thr Ala Thr Val Leu Asp Ser
275 280 285
Asp Gly Ser Tyr Phe Met Tyr Ser Lys Leu Arg Val Gln Lys Ser Thr
290 295 300
Trp Glu Arg Gly Ser Leu Phe Ala Cys Ser Val Val His Glu Val Leu
305 310 315 320
His Asn His Leu Thr Thr Lys Thr Ile Ser Arg Ser Leu Gly Lys
325 330 335
<210> 57
<211> 1008
<212> ДНК
<213> Bubalus arnee
<400> 57
gccaaaacaa cagccccatc ggtctatcca ctggcccctg tgtgtggagg tacaactggc 60
tcctcggtga ctctaggatg cctggtcaag ggttatttcc ctgagccagt gaccttgacc 120
tggaactctg gatccctgtc cagtggtgtg cacaccttcc cagctctcct gcagtctggc 180
ctctacaccc tcagcagctc agtgactgta acctcgaaca cctggcccag ccagaccatc 240
acctgcaatg tggcccaccc ggcaagcagc accaaagtgg acaagaaaat tgagcccaga 300
gtgcccataa cacagaaccc ctgtcctcca ctcaaagagt gtcccccatg cgcagctcca 360
gacctcttgg gtggaccatc cgtcttcatc ttccctccaa agatcaagga tgtactcatg 420
atctccctga gccccatggt cacatgtgtg gtggtggatg tgagcgagga tgacccagac 480
gtccagatca gctggtttgt gaacaacgtg gaagtacaca cagctcagac acaaacccat 540
agagaggatt acaacagtac tctccgggtg gtcagtgccc tccccatcca gcaccaggac 600
tggatgagtg gcaaggagtt caaatgcaag gtcaacaaca gagccctccc atcccccatc 660
gagaaaacca tctcaaaacc cagagggcca gtaagagctc cacaggtata tgtcttgcct 720
ccaccagcag aagagatgac taagaaagag ttcagtctga cctgcatgat cacaggcttc 780
ttacctgccg aaattgctgt ggactggacc agcaatgggc gtacagagca aaactacaag 840
aacaccgcaa cagtcctgga ctctgatggt tcttacttca tgtacagcaa gctcagagta 900
caaaagagca cttgggaaag aggaagtctt ttcgcctgct cagtggtcca cgaggtgctg 960
cacaatcacc ttacgactaa gaccatctcc cggtctctgg gtaaatga 1008
<210> 58
<211> 335
<212> Белок
<213> Bubalus arnee
<400> 58
Ala Lys Thr Thr Ala Pro Ser Val Tyr Pro Leu Ala Pro Val Cys Gly
1 5 10 15
Gly Thr Thr Gly Ser Ser Val Thr Leu Gly Cys Leu Val Lys Gly Tyr
20 25 30
Phe Pro Glu Pro Val Thr Leu Thr Trp Asn Ser Gly Ser Leu Ser Ser
35 40 45
Gly Val His Thr Phe Pro Ala Leu Leu Gln Ser Gly Leu Tyr Thr Leu
50 55 60
Ser Ser Ser Val Thr Val Thr Ser Asn Thr Trp Pro Ser Gln Thr Ile
65 70 75 80
Thr Cys Asn Val Ala His Pro Ala Ser Ser Thr Lys Val Asp Lys Lys
85 90 95
Ile Glu Ser Arg Arg Pro Ile Pro Pro Asn Ser Cys Pro Pro Cys Lys
100 105 110
Glu Cys Ser Ile Phe Pro Ala Pro Asp Leu Leu Gly Gly Pro Ser Val
115 120 125
Phe Ile Phe Pro Pro Lys Ile Lys Asp Val Leu Met Ile Ser Leu Ser
130 135 140
Pro Ile Val Thr Cys Val Val Val Asp Val Ser Glu Asp Asp Pro Asp
145 150 155 160
Val Gln Ile Ser Trp Phe Val Asn Asn Val Glu Val His Thr Ala Gln
165 170 175
Thr Gln Thr His Arg Glu Asp Tyr Asn Ser Thr Leu Arg Val Val Ser
180 185 190
Ala Leu Pro Ile Gln His Gln Asp Trp Met Ser Gly Lys Glu Phe Lys
195 200 205
Cys Lys Val Asn Asn Arg Ala Leu Pro Ser Pro Ile Glu Lys Thr Ile
210 215 220
Ser Lys Pro Arg Gly Pro Val Arg Ala Pro Gln Val Tyr Val Leu Pro
225 230 235 240
Pro Pro Ala Glu Glu Met Thr Lys Lys Glu Phe Ser Leu Thr Cys Met
245 250 255
Ile Thr Asp Phe Leu Pro Ala Glu Ile Ala Val Asp Trp Thr Ser Asn
260 265 270
Gly His Lys Glu Leu Asn Tyr Lys Asn Thr Ala Pro Val Leu Asp Thr
275 280 285
Asp Gly Ser Tyr Phe Met Tyr Ser Lys Leu Arg Val Gln Lys Ser Thr
290 295 300
Trp Glu Lys Gly Ser Leu Phe Ala Cys Ser Val Val His Glu Gly Leu
305 310 315 320
His Asn His His Thr Thr Lys Thr Ile Ser Arg Ser Leu Gly Lys
325 330 335
<210> 59
<211> 1008
<212> ДНК
<213> Bubalus arnee
<400> 59
gccaaaacaa cagccccatc ggtctatcca ctggcccctg tgtgtggagg tacaactggc 60
tcctcggtga ctctaggatg cctggtcaag ggttatttcc ctgagccagt gaccttgacc 120
tggaactctg gatccctgtc cagtggtgtg cacaccttcc cagctctcct gcagtctggc 180
ctctacaccc tcagcagctc agtgactgta acctcgaaca cctggcccag ccagaccatc 240
acctgcaatg tggcccaccc ggcaagcagc accaaagtgg acaagaaaat tgaatccaga 300
aggcccatac cacccaactc ctgtcctcca tgcaaagagt gttccatatt cccagctcct 360
gacctcttgg gtggaccatc cgtcttcatc ttccctccaa agatcaagga tgtactcatg 420
atctccctga gccccatagt cacatgtgtg gtggtggatg tgagcgagga tgacccagat 480
gtccagatca gctggtttgt gaacaacgtg gaagtacaca cagctcagac acaaacccat 540
agagaggatt acaacagtac tctccgggtg gtcagtgccc tccccatcca gcaccaggac 600
tggatgagtg gcaaggagtt caaatgcaag gtcaacaaca gagccctccc atcccccatc 660
gagaaaacca tctcaaaacc cagagggcca gtaagagctc cacaggtata tgtcttgcct 720
ccaccagcag aagagatgac taagaaagag ttcagtctga cctgcatgat cacagacttc 780
ttacctgccg aaattgctgt ggactggacc agcaatgggc ataaagagct gaactacaag 840
aacaccgcac cagtcctgga cactgatggt tcttacttca tgtacagcaa gctcagagtg 900
caaaagagca cttgggaaaa aggaagtctt ttcgcctgct cagtggtcca cgagggtctg 960
cacaatcacc atacgactaa gaccatctcc cggtctctgg gtaaatga 1008
<210> 60
<211> 335
<212> Белок
<213> Bubalus arnee
<400> 60
Ala Lys Thr Thr Ala Pro Ser Val Tyr Pro Leu Ala Pro Val Cys Gly
1 5 10 15
Gly Thr Thr Gly Ser Ser Val Thr Leu Gly Cys Leu Val Lys Gly Tyr
20 25 30
Phe Pro Glu Pro Val Thr Leu Thr Trp Asn Ser Gly Ser Leu Ser Ser
35 40 45
Gly Val His Thr Phe Pro Ala Leu Leu Gln Ser Gly Leu Tyr Thr Leu
50 55 60
Ser Ser Ser Val Thr Val Thr Ser Asn Thr Trp Pro Ser Gln Thr Ile
65 70 75 80
Thr Cys Asn Val Ala His Pro Ala Ser Ser Thr Lys Val Asp Lys Lys
85 90 95
Ile Glu Pro Arg Val Pro Ile Thr Gln Asn Pro Cys Pro Pro Leu Lys
100 105 110
Glu Cys Pro Pro Cys Ala Ala Pro Asp Leu Leu Gly Gly Pro Ser Val
115 120 125
Phe Ile Phe Pro Pro Lys Ile Lys Asp Val Leu Met Ile Ser Leu Ser
130 135 140
Pro Met Val Thr Cys Val Val Val Asp Val Ser Glu Asp Asp Pro Asp
145 150 155 160
Val Gln Ile Ser Trp Phe Val Asn Asn Val Glu Val His Thr Ala Gln
165 170 175
Thr Gln Thr His Arg Glu Asp Tyr Asn Ser Thr Leu Arg Val Val Ser
180 185 190
Ala Leu Pro Ile Gln His Gln Asp Trp Met Ser Gly Lys Glu Phe Lys
195 200 205
Cys Lys Val Asn Asn Arg Ala Leu Pro Ser Pro Ile Glu Lys Thr Ile
210 215 220
Ser Lys Pro Arg Gly Pro Val Arg Ala Pro Gln Val Tyr Val Leu Pro
225 230 235 240
Pro Pro Ala Glu Glu Met Thr Lys Lys Glu Phe Ser Leu Thr Cys Met
245 250 255
Ile Thr Gly Phe Leu Pro Ala Glu Ile Ala Val Asp Trp Thr Ser Asn
260 265 270
Gly Arg Thr Glu Gln Asn Tyr Lys Asn Thr Ala Thr Val Leu Asp Ser
275 280 285
Asp Gly Ser Tyr Phe Met Tyr Ser Lys Leu Arg Val Gln Lys Ser Thr
290 295 300
Trp Glu Arg Gly Ser Leu Phe Ala Cys Ser Val Val His Glu Gly Leu
305 310 315 320
His Asn His Leu Thr Thr Lys Thr Ile Ser Arg Ser Leu Gly Lys
325 330 335
<210> 61
<211> 1008
<212> ДНК
<213> Bubalus arnee
<400> 61
gccaaaacaa cagccccatc ggtctatcca ctggcccctg tgtgtggagg tacaactggc 60
tcctcggtga ctctaggatg cctggtcaag ggttatttcc ctgagccagt gaccttgacc 120
tggaactctg gatccctgtc cagtggtgtg cacaccttcc cagctctcct gcagtctggc 180
ctctacaccc tcagcagctc agtgactgta acctcgaaca cctggcccag ccagaccatc 240
acctgcaatg tggcccaccc ggcaagcagc accaaagtgg acaagaaaat tgagcccaga 300
gtgcccataa cacagaaccc ctgtcctcca ctcaaagagt gtcccccatg cgcagctcca 360
gacctcttgg gtggaccatc cgtcttcatc ttccctccaa agatcaagga tgtactcatg 420
atctccctga gccccatggt cacatgtgtg gtggtggatg tgagcgagga tgacccagac 480
gtccagatca gctggtttgt gaacaacgtg gaagtacaca cagctcagac acaaacccat 540
agagaggatt acaacagtac tctccgggtg gtcagtgccc tccccatcca gcaccaggac 600
tggatgagtg gcaaggagtt caaatgcaag gtcaacaaca gagccctccc atcccccatc 660
gagaaaacca tctcaaaacc cagagggcca gtaagagctc cacaggtata tgtcttgcct 720
ccaccagcag aagagatgac taagaaagag ttcagtctga cctgcatgat cacaggcttc 780
ttacctgccg aaattgctgt ggactggacc agcaatgggc gtacagagca aaactacaag 840
aacaccgcaa cagtcctgga ctctgatggt tcttacttca tgtacagcaa gctcagagta 900
caaaagagca cttgggaaag aggaagtctt ttcgcctgct cagtggtcca cgagggtctg 960
cacaatcacc ttacgactaa gaccatctcc cggtctctgg gtaaatga 1008
<210> 62
<211> 330
<212> Белок
<213> Bubalus arnee
<400> 62
Ala Thr Thr Thr Ala Pro Ser Val Tyr Pro Leu Val Pro Gly Cys Ser
1 5 10 15
Asp Thr Ser Gly Ser Ser Val Thr Leu Gly Cys Leu Val Lys Gly Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Lys Trp Asn Tyr Gly Ala Leu Ser Ser
35 40 45
Gly Val Arg Thr Val Ser Ser Val Leu Gln Ser Gly Phe Tyr Ser Leu
50 55 60
Ser Ser Leu Val Thr Val Pro Ser Ser Thr Trp Pro Ser Gln Thr Val
65 70 75 80
Ile Cys Asn Val Ala His Pro Ala Ser Lys Thr Glu Leu Ile Lys Arg
85 90 95
Ile Glu Pro Arg Ile Pro Lys Pro Ser Thr Pro Pro Gly Ser Ser Cys
100 105 110
Pro Pro Gly Asn Ile Leu Gly Gly Pro Ser Val Phe Ile Phe Pro Pro
115 120 125
Lys Pro Lys Asp Ala Leu Met Ile Ser Leu Thr Pro Lys Val Thr Cys
130 135 140
Val Val Val Asp Val Ser Glu Asp Asp Pro Asp Val His Val Ser Trp
145 150 155 160
Phe Val Asp Asn Lys Glu Val His Thr Ala Trp Thr Gln Pro Arg Glu
165 170 175
Ala Gln Tyr Asn Ser Thr Phe Arg Val Val Ser Ala Leu Pro Ile Gln
180 185 190
His Gln Asp Trp Met Arg Gly Lys Glu Phe Lys Cys Lys Val Asn Asn
195 200 205
Lys Ala Leu Pro Ala Pro Ile Glu Arg Thr Ile Ser Lys Pro Lys Gly
210 215 220
Arg Ala Gln Thr Pro Gln Val Tyr Thr Ile Pro Pro Pro Arg Glu Gln
225 230 235 240
Met Ser Lys Lys Lys Val Ser Leu Thr Cys Leu Val Thr Asn Phe Phe
245 250 255
Ser Glu Ala Ile Ser Val Glu Trp Glu Arg Asn Gly Glu Leu Glu Gln
260 265 270
Asp Tyr Lys Asn Thr Pro Pro Ile Leu Asp Ser Asp Gly Thr Tyr Phe
275 280 285
Leu Tyr Ser Lys Leu Thr Val Asp Thr Asp Ser Trp Leu Gln Gly Glu
290 295 300
Ile Phe Thr Cys Ser Val Val His Glu Ala Leu His Asn His His Thr
305 310 315 320
Gln Lys Asn Leu Ser Arg Ser Pro Gly Lys
325 330
<210> 63
<211> 993
<212> ДНК
<213> Bubalus arnee
<400> 63
gctacaacaa cagccccatc tgtctatccc ttggtccctg gctgcagtga cacatctgga 60
tcctcggtga cactgggatg ccttgtcaaa ggctacttcc ctgagccggt aactgtaaaa 120
tggaactatg gagccctgtc cagcggtgtg cgcacagtct catctgtcct gcagtctggg 180
ttctattccc tcagcagctt ggtgactgta ccctccagca cctggcccag ccagactgtc 240
atctgcaacg tagcccaccc agccagcaag actgagttga tcaagagaat cgagcctaga 300
atacccaagc ccagtacccc cccaggttct tcatgcccac ctggtaacat cttgggtgga 360
ccatccgtct tcatcttccc cccaaagccc aaggatgcac tcatgatctc cctaaccccc 420
aaggttacgt gtgtggtggt ggatgtgagc gaggatgacc cagatgtcca tgtcagctgg 480
tttgtggaca acaaagaagt acacacagcc tggacacagc cccgtgaagc tcagtacaac 540
agtaccttcc gagtggtcag tgccctcccc atccagcacc aggactggat gaggggcaag 600
gagttcaaat gcaaggtcaa caacaaagcc ctcccagccc ccatcgagag aaccatctca 660
aaacccaaag gaagagccca gacacctcaa gtatacacca tacccccacc tcgtgaacaa 720
atgtccaaga agaaggttag tctgacctgc ctggtcacca acttcttctc tgaagccatc 780
agtgtggagt gggaaaggaa cggagaactg gagcaggatt acaagaacac tccacccatc 840
ctggactcag atgggaccta cttcctctac agcaagctca ctgtggatac agacagttgg 900
ttgcaaggag aaatttttac ctgctccgtg gtgcatgagg ctctccataa ccaccacaca 960
cagaagaacc tgtctcgctc ccctggtaaa tga 993
<210> 64
<211> 105
<212> Белок
<213> Bubalus arnee
<400> 64
Gln Pro Lys Ser Pro Pro Ser Val Thr Leu Phe Pro Pro Ser Thr Glu
1 5 10 15
Glu Leu Asn Gly Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe
20 25 30
Tyr Pro Gly Ser Val Thr Val Val Trp Lys Ala Asp Gly Ser Thr Ile
35 40 45
Thr Arg Asn Val Glu Thr Thr Arg Ala Ser Lys Gln Ser Asn Ser Lys
50 55 60
Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Ser Ser Asp Trp Lys Ser
65 70 75 80
Lys Gly Ser Tyr Ser Cys Glu Val Thr His Glu Gly Ser Thr Val Thr
85 90 95
Lys Thr Val Lys Pro Ser Glu Cys Ser
100 105
<210> 65
<211> 318
<212> ДНК
<213> Bubalus arnee
<400> 65
cagcccaagt ccccaccctc ggtcaccctg ttcccgccct ccacggagga gctcaacggc 60
aacaaggcca ccctggtgtg tctcatcagc gacttctacc cgggtagcgt gaccgtggtc 120
tggaaggcag acggcagcac catcacccgc aacgtggaga ccacccgggc ctccaaacag 180
agcaacagca agtacgcggc cagcagctac ctgagcctga cgagcagcga ctggaaatcg 240
aaaggcagtt acagctgcga ggtcacgcac gaggggagca ccgtgacgaa gacagtgaag 300
ccctcagagt gttcttag 318
<210> 66
<211> 329
<212> Белок
<213> Bubalus arnee
<400> 66
Ala Ser Thr Thr Ala Pro Lys Val Tyr Pro Leu Ser Ser Cys Cys Gly
1 5 10 15
Asp Lys Ser Ser Ser Thr Val Thr Leu Gly Cys Leu Val Ser Ser Tyr
20 25 30
Met Pro Glu Pro Val Thr Val Thr Trp Asn Ser Gly Ala Leu Lys Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Met Val Thr Val Pro Gly Ser Thr Ser Gly Gln Thr Phe
65 70 75 80
Thr Cys Asn Val Ala His Pro Ala Ser Ser Thr Lys Val Asp Lys Ala
85 90 95
Val Asp Pro Thr Cys Lys Pro Ser Pro Cys Asp Cys Cys Pro Pro Pro
100 105 110
Glu Leu Pro Gly Gly Pro Ser Val Phe Ile Phe Pro Pro Lys Pro Lys
115 120 125
Asp Thr Leu Thr Ile Ser Gly Thr Pro Glu Val Thr Cys Val Val Val
130 135 140
Asp Val Gly His Asp Asp Pro Glu Val Lys Phe Ser Trp Phe Val Asp
145 150 155 160
Asp Val Glu Val Asn Thr Ala Thr Thr Lys Pro Arg Glu Glu Gln Phe
165 170 175
Asn Ser Thr Tyr Arg Val Val Ser Ala Leu Arg Ile Gln His Gln Asp
180 185 190
Trp Thr Gly Gly Lys Glu Phe Lys Cys Lys Val His Asn Glu Gly Leu
195 200 205
Pro Ala Pro Ile Val Arg Thr Ile Ser Arg Thr Lys Gly Pro Ala Arg
210 215 220
Glu Pro Gln Val Tyr Val Leu Ala Pro Pro Gln Glu Glu Leu Ser Lys
225 230 235 240
Ser Thr Val Ser Leu Thr Cys Met Val Thr Ser Phe Tyr Pro Asp Tyr
245 250 255
Ile Ala Val Glu Trp Gln Arg Asn Gly Gln Pro Glu Ser Glu Asp Lys
260 265 270
Tyr Gly Thr Thr Pro Pro Gln Leu Asp Ala Asp Ser Ser Tyr Phe Leu
275 280 285
Tyr Ser Lys Leu Arg Val Asp Arg Asn Ser Trp Gln Glu Gly Asp Thr
290 295 300
Tyr Thr Cys Val Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
305 310 315 320
Lys Ser Thr Ser Lys Ser Ala Gly Lys
325
<210> 67
<211> 990
<212> ДНК
<213> Bubalus arnee
<400> 67
gcctccacca cagccccgaa agtctaccct ctgagttctt gctgcgggga caagtccagc 60
tccaccgtga ccctgggctg cctggtctcc agctacatgc ccgagccggt gaccgtgacc 120
tggaactcgg gtgccctgaa gagcggcgtg cacaccttcc cggctgtcct tcagtcctcc 180
gggctgtact ctctcagcag catggtgacc gtgcccggca gcacctcagg acagaccttc 240
acctgcaacg tagcccaccc ggccagcagc accaaggtgg acaaggctgt tgatcccaca 300
tgcaaaccat caccctgtga ctgttgccca ccccctgagc tccccggagg accctctgtc 360
ttcatcttcc caccgaaacc caaggacacc ctcacaatct cgggaacgcc cgaggtcacg 420
tgtgtggtgg tggacgtggg ccacgatgac cccgaggtga agttctcctg gttcgtggac 480
gacgtggagg taaacacagc cacgacgaag ccgagagagg agcagttcaa cagcacctac 540
cgcgtggtca gcgccctgcg catccagcac caggactgga ctggaggaaa ggagttcaag 600
tgcaaggtcc acaacgaagg cctcccggcc cccatcgtga ggaccatctc caggaccaaa 660
gggccggccc gggagccgca ggtgtatgtc ctggccccac cccaggaaga gctcagcaaa 720
agcacggtca gcctcacctg catggtcacc agcttctacc cagactacat cgccgtggag 780
tggcagagaa acgggcagcc tgagtcggag gacaagtacg gcacgacccc gccccagctg 840
gacgccgaca gctcctactt cctgtacagc aagctcaggg tggacaggaa cagctggcag 900
gaaggagaca cctacacgtg tgtggtgatg cacgaggccc tgcacaatca ctacacgcag 960
aagtccacct ctaagtctgc gggtaaatga 990
<210> 68
<211> 329
<212> Белок
<213> Bubalus arnee
<400> 68
Ala Ser Thr Thr Ala Pro Lys Val Tyr Pro Leu Ser Ser Cys Cys Gly
1 5 10 15
Asp Lys Ser Ser Ser Thr Val Thr Leu Gly Cys Leu Val Ser Ser Tyr
20 25 30
Met Pro Glu Pro Val Thr Val Thr Trp Asn Ser Gly Ala Leu Lys Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Met Val Thr Val Pro Gly Ser Thr Ser Gly Gln Thr Phe
65 70 75 80
Thr Cys Asn Val Ala His Pro Ala Ser Ser Thr Lys Val Asp Lys Ala
85 90 95
Val Asp Pro Thr Cys Lys Pro Ser Pro Cys Asp Cys Cys Pro Pro Pro
100 105 110
Glu Leu Pro Gly Gly Pro Ser Val Phe Ile Phe Pro Pro Lys Pro Lys
115 120 125
Asp Thr Leu Thr Ile Ser Gly Thr Pro Glu Val Thr Cys Val Val Val
130 135 140
Asp Val Gly His Asp Asp Pro Glu Val Lys Phe Ser Trp Phe Val Asp
145 150 155 160
Asp Val Glu Val Asn Thr Ala Thr Thr Lys Pro Arg Glu Glu Gln Phe
165 170 175
Asn Ser Thr Tyr Arg Val Val Ser Ala Leu Arg Ile Gln His Gln Asp
180 185 190
Trp Thr Gly Gly Lys Glu Phe Lys Cys Lys Val His Asn Glu Gly Leu
195 200 205
Pro Ala Pro Ile Val Arg Thr Ile Ser Arg Thr Lys Gly Pro Ala Arg
210 215 220
Glu Pro Gln Val Tyr Val Leu Ala Pro Pro Gln Glu Glu Leu Ser Lys
225 230 235 240
Ser Thr Val Ser Leu Thr Cys Met Val Thr Ser Phe Tyr Pro Asp Tyr
245 250 255
Ile Ala Val Glu Trp Gln Arg Asn Gly Gln Pro Glu Ser Glu Asp Lys
260 265 270
Tyr Gly Thr Thr Pro Pro Gln Leu Asp Ala Asp Ser Ser Tyr Phe Leu
275 280 285
Tyr Ser Lys Leu Arg Val Asp Arg Asn Ser Trp Gln Glu Gly Asp Thr
290 295 300
Tyr Thr Cys Val Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
305 310 315 320
Lys Ser Thr Ser Lys Ser Ala Gly Lys
325
<210> 69
<211> 990
<212> ДНК
<213> Bubalus arnee
<400> 69
gcctccacca cagccccgaa agtctaccct ctgagttctt gctgcgggga caagtccagc 60
tccaccgtga ccctgggctg cctggtctcc agctacatgc ccgagccggt gaccgtgacc 120
tggaactcgg gtgccctgaa gagcggcgtg cacaccttcc cggccgtcct tcagtcctcc 180
gggctgtact ctctcagcag catggtgacc gtgcccggca gcacctcagg acagaccttc 240
acctgcaacg tagcccaccc ggccagcagc accaaggtgg acaaggctgt tgatcccaca 300
tgcaaaccat caccctgtga ctgttgccca ccccctgagc tccccggagg accctctgtc 360
ttcatcttcc caccgaaacc caaggacacc ctcacaatct cgggaacgcc cgaggtcacg 420
tgtgtggtgg tggacgtggg ccacgatgac cccgaggtga agttctcctg gttcgtggac 480
gacgtggagg taaacacagc cacgacgaag ccgagagagg agcagttcaa cagcacctac 540
cgcgtggtca gcgccctgcg catccagcac caggactgga ctggaggaaa ggagttcaag 600
tgcaaggtcc acaacgaagg cctcccggcc cccatcgtga ggaccatctc caggaccaaa 660
gggccggccc gggagccgca ggtgtatgtc ctggccccac cccaggaaga gctcagcaaa 720
agcacggtca gcctcacctg catggtcacc agcttctacc cagactacat cgccgtggag 780
tggcagagaa acgggcagcc tgagtcggag gacaagtacg gcacgacccc gccccagctg 840
gacgccgaca gctcctactt cctgtacagc aagctcaggg tggacaggaa cagctggcag 900
gaaggagaca cctacacgtg tgtggtgatg cacgaggccc tgcacaatca ctacacgcag 960
aagtccacct ctaagtctgc gggtaaatga 990
<210> 70
<211> 329
<212> Белок
<213> Bubalus arnee
<400> 70
Ala Ser Thr Thr Ala Pro Lys Val Tyr Pro Leu Ser Ser Cys Cys Gly
1 5 10 15
Asp Lys Ser Ser Ser Thr Val Thr Leu Gly Cys Leu Val Ser Ser Tyr
20 25 30
Met Pro Glu Pro Val Thr Val Thr Trp Asn Ser Gly Ala Leu Lys Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Met Val Thr Val Pro Gly Ser Thr Ser Gly Thr Gln Thr
65 70 75 80
Phe Thr Cys Asn Val Ala His Pro Ala Ser Ser Thr Lys Val Asp Lys
85 90 95
Ala Val Asp Pro Arg Cys Lys Thr Thr Cys Asp Cys Cys Pro Pro Pro
100 105 110
Glu Leu Pro Gly Gly Pro Ser Val Phe Ile Phe Pro Pro Lys Pro Lys
115 120 125
Asp Thr Leu Thr Ile Ser Gly Thr Pro Glu Val Thr Cys Val Val Val
130 135 140
Asp Val Gly His Asp Asp Pro Glu Val Lys Phe Ser Trp Phe Val Asp
145 150 155 160
Asp Val Glu Val Asn Thr Ala Thr Thr Lys Pro Arg Glu Glu Gln Phe
165 170 175
Asn Ser Thr Tyr Arg Val Val Ser Ala Leu Arg Ile Gln His Gln Asp
180 185 190
Trp Thr Gly Gly Lys Glu Phe Lys Cys Lys Val His Asn Glu Gly Leu
195 200 205
Pro Ala Pro Ile Val Arg Thr Ile Ser Arg Thr Lys Gly Pro Ala Arg
210 215 220
Glu Pro Gln Val Tyr Val Leu Ala Pro Pro Gln Glu Glu Leu Ser Lys
225 230 235 240
Ser Thr Val Ser Leu Thr Cys Met Val Thr Ser Phe Tyr Pro Asp Tyr
245 250 255
Ile Ala Val Glu Trp Gln Arg Asn Gly Gln Pro Glu Ser Glu Asp Lys
260 265 270
Tyr Gly Thr Thr Pro Pro Gln Leu Asp Ala Asp Gly Ser Tyr Phe Leu
275 280 285
Tyr Ser Arg Leu Arg Val Asp Arg Asn Ser Trp Gln Glu Gly Asp Thr
290 295 300
Tyr Thr Cys Val Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
305 310 315 320
Lys Ser Thr Ser Lys Ser Ala Gly Lys
325
<210> 71
<211> 990
<212> ДНК
<213> Bubalus arnee
<400> 71
gcctccacca cagccccgaa agtctaccct ctgagttctt gctgcgggga caagtccagc 60
tccaccgtga ccctgggctg cctggtctcc agctacatgc ccgagccggt gaccgtgacc 120
tggaactcgg gtgccctgaa gagcggcgtg cacaccttcc cggccgtcct tcagtcctcc 180
gggctctact ctctcagcag catggtgacc gtgcccggca gcacctcagg aacccagacc 240
ttcacctgca acgtagccca cccggccagc agcaccaagg tggacaaggc tgttgatccc 300
agatgcaaaa caacctgtga ctgttgccca ccgcctgagc tccctggagg accctctgtc 360
ttcatcttcc caccgaaacc caaggacacc ctcacaatct cgggaacgcc cgaggtcacg 420
tgtgtggtgg tggacgtggg ccacgatgac cccgaggtga agttctcctg gttcgtggac 480
gacgtggagg taaacacagc cacgacgaag ccgagagagg agcagttcaa cagcacctac 540
cgcgtggtca gcgccctgcg catccagcac caggactgga ctggaggaaa ggagttcaag 600
tgcaaggtcc acaacgaagg cctcccagcc cccatcgtga ggaccatctc caggaccaaa 660
gggccggccc gggagccgca ggtgtatgtc ctggccccac cccaggaaga gctcagcaaa 720
agcacggtca gcctcacctg catggtcacc agcttctacc cagactacat cgccgtggag 780
tggcagagaa atgggcagcc tgagtcagag gacaagtacg gcacgacccc tccccagctg 840
gacgccgacg gctcctactt cctgtacagc aggctcaggg tggacaggaa cagctggcag 900
gaaggagaca cctacacgtg tgtggtgatg cacgaggccc tgcacaatca ctacacgcag 960
aagtccacct ctaagtctgc gggtaaatga 990
<210> 72
<211> 326
<212> Белок
<213> Bubalus arnee
<400> 72
Ala Ser Thr Thr Ala Pro Lys Val Tyr Pro Leu Ala Ser Ser Cys Gly
1 5 10 15
Asp Thr Ser Ser Ser Thr Val Thr Leu Gly Cys Leu Val Ser Ser Tyr
20 25 30
Met Pro Glu Pro Val Thr Val Thr Trp Asn Ser Gly Ala Leu Lys Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Met Val Thr Val Pro Ala Ser Ser Ser Gly Gln Thr Phe
65 70 75 80
Thr Cys Asn Val Ala His Pro Ala Ser Ser Thr Lys Val Asp Lys Ala
85 90 95
Val Gly Val Ser Ile Asp Cys Ser Lys Cys His Asn Gln Pro Cys Val
100 105 110
Arg Glu Pro Ser Val Phe Ile Phe Pro Pro Lys Pro Lys Asp Thr Leu
115 120 125
Met Ile Thr Gly Thr Pro Glu Val Thr Cys Val Val Val Asn Val Gly
130 135 140
His Asp Asn Pro Glu Val Gln Phe Ser Trp Phe Val Asp Asp Val Glu
145 150 155 160
Val His Thr Ala Arg Ser Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr
165 170 175
Tyr Arg Val Val Ser Ala Leu Pro Ile Gln His Gln Asp Trp Thr Gly
180 185 190
Gly Lys Glu Phe Lys Cys Lys Val Asn Asn Lys Gly Leu Ser Ala Pro
195 200 205
Ile Val Arg Ile Ile Ser Arg Ser Lys Gly Pro Ala Arg Glu Pro Gln
210 215 220
Val Tyr Val Leu Asp Pro Pro Lys Glu Glu Leu Ser Lys Ser Thr Leu
225 230 235 240
Ser Val Thr Cys Met Val Thr Gly Phe Tyr Pro Glu Asp Val Ala Val
245 250 255
Glu Trp Gln Arg Asn Arg Gln Thr Glu Ser Glu Asp Lys Tyr Arg Thr
260 265 270
Thr Pro Pro Gln Leu Asp Thr Asp Arg Ser Tyr Phe Leu Tyr Ser Lys
275 280 285
Leu Arg Val Asp Arg Asn Ser Trp Gln Glu Gly Asp Ala Tyr Thr Cys
290 295 300
Val Val Met His Glu Ala Leu His Asn His Tyr Met Gln Lys Ser Thr
305 310 315 320
Ser Lys Ser Ala Gly Lys
325
<210> 73
<211> 981
<212> ДНК
<213> Bubalus arnee
<400> 73
gcctccacca cagccccgaa agtctaccct ctggcatcca gctgcggaga cacatccagc 60
tccaccgtga ccctgggctg cctggtgtcc agctacatgc ccgagccggt gaccgtgacc 120
tggaactcgg gtgccctgaa gagcggcgtg cacaccttcc cggctgtcct tcagtcctcc 180
gggctctact ctctcagcag catggtgacc gtgcccgcca gcagctcagg acagaccttc 240
acctgcaacg tagcccaccc ggccagcagc accaaggtgg acaaggctgt tggggtctcc 300
attgactgct ccaagtgtca taaccagcct tgcgtgaggg aaccatctgt cttcatcttc 360
ccaccgaaac ccaaagacac cctgatgatc acaggaacgc ccgaggtcac gtgtgtggtg 420
gtgaacgtgg gccacgataa ccccgaggtg cagttctcct ggttcgtgga tgacgtggag 480
gtgcacacgg ccaggtcgaa gccaagagag gagcagttca acagcacgta ccgcgtggtc 540
agcgccctgc ccatccagca ccaggactgg actggaggaa aggagttcaa gtgcaaggtc 600
aacaacaaag gcctctcggc ccccatcgtg aggatcatct ccaggagcaa agggccggcc 660
cgggagccgc aggtgtatgt cctggaccca cccaaggaag agctcagcaa aagcacgctc 720
agcgtcacct gcatggtcac cggcttctac ccagaagatg tagccgtgga gtggcagaga 780
aaccggcaga ctgagtcgga ggacaagtac cgcacgaccc cgccccagct ggacaccgac 840
cgctcctact tcctgtacag caagctcagg gtggacagga acagctggca ggaaggagac 900
gcctacacgt gtgtggtgat gcacgaggcc ctgcacaatc actacatgca gaagtccacc 960
tctaagtctg cgggtaaatg a 981
<210> 74
<211> 326
<212> Белок
<213> Bubalus arnee
<400> 74
Ala Ser Thr Thr Ala Pro Lys Val Tyr Pro Leu Ser Ser Cys Cys Gly
1 5 10 15
Asp Lys Ser Ser Ser Thr Val Thr Leu Gly Cys Leu Val Ser Ser Tyr
20 25 30
Met Pro Glu Pro Val Thr Val Thr Trp Asn Ser Gly Ala Leu Lys Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Met Val Thr Val Pro Gly Ser Thr Ser Gly Gln Thr Phe
65 70 75 80
Thr Cys Asn Val Ala His Pro Ala Ser Ser Thr Lys Val Asp Lys Ala
85 90 95
Val Gly Val Ser Ser Asp Cys Ser Lys Pro Asn Asn Gln His Cys Val
100 105 110
Arg Glu Pro Ser Val Phe Ile Phe Pro Pro Lys Pro Lys Asp Thr Leu
115 120 125
Met Ile Thr Gly Thr Pro Glu Val Thr Cys Val Val Val Asn Val Gly
130 135 140
His Asp Asn Pro Glu Val Gln Phe Ser Trp Phe Val Asp Asp Val Glu
145 150 155 160
Val His Thr Ala Arg Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr
165 170 175
Tyr Arg Val Val Ser Ala Leu Pro Ile Gln His Gln Asp Trp Thr Gly
180 185 190
Gly Lys Glu Phe Lys Cys Lys Val Asn Ile Lys Gly Leu Ser Ala Ser
195 200 205
Ile Val Arg Ile Ile Ser Arg Ser Lys Gly Pro Ala Arg Glu Pro Gln
210 215 220
Val Tyr Val Leu Asp Pro Pro Lys Glu Glu Leu Ser Lys Ser Thr Val
225 230 235 240
Ser Val Thr Cys Met Val Ile Gly Phe Tyr Pro Glu Asp Val Asp Val
245 250 255
Glu Trp Gln Arg Asp Arg Gln Thr Glu Ser Glu Asp Lys Tyr Arg Thr
260 265 270
Thr Pro Pro Gln Leu Asp Ala Asp Arg Ser Tyr Phe Leu Tyr Ser Lys
275 280 285
Leu Arg Val Asp Arg Asn Ser Trp Gln Arg Gly Asp Thr Tyr Thr Cys
290 295 300
Val Val Met His Glu Ala Leu His Asn His Tyr Met Gln Lys Ser Thr
305 310 315 320
Ser Lys Ser Ala Gly Lys
325
<210> 75
<211> 981
<212> ДНК
<213> Bubalus arnee
<400> 75
gcctccacca cagccccgaa agtctaccct ctgagttctt gctgcgggga caagtccagc 60
tccaccgtga ccctgggctg cctggtgtcc agctacatgc ccgagccggt gaccgtgacc 120
tggaactcgg gtgccctgaa gagcggcgtg cacaccttcc cggccgtcct tcagtcctcc 180
gggctctact ctctcagcag catggtgacc gtgcccggca gcacctcagg acagaccttc 240
acctgcaacg tagcccaccc ggccagcagc accaaggtgg acaaggctgt tggggtctcc 300
agtgactgct ccaagcctaa taaccagcat tgcgtgaggg aaccatctgt cttcatcttc 360
ccaccgaaac ccaaagacac cctgatgatc acaggaacgc ccgaggtcac gtgtgtggtg 420
gtgaacgtgg gccacgataa ccccgaggtg cagttctcct ggttcgtgga cgacgtggag 480
gtgcacacgg ccaggacgaa gccgagagag gagcagttca acagcacgta ccgcgtggtc 540
agcgccctgc ccatccagca ccaggactgg actggaggaa aggagttcaa gtgcaaggtc 600
aacatcaaag gcctctcggc ctccatcgtg aggatcatct ccaggagcaa agggccggcc 660
cgggagccgc aggtgtatgt cctggaccca cccaaggaag agctcagcaa aagcacggtc 720
agcgtcacct gcatggtcat cggcttctac ccagaagatg tagacgtgga gtggcagaga 780
gaccggcaga ctgagtcgga ggacaagtac cgcacgaccc cgccccagct ggacgccgac 840
cgctcctact tcctgtacag caagctcagg gtggacagga acagctggca gagaggagac 900
acctacacgt gtgtggtgat gcacgaggcc ctgcacaatc actacatgca gaagtccacc 960
tctaagtctg cgggtaaatg a 981
<210> 76
<211> 327
<212> Белок
<213> Bubalus arnee
<400> 76
Ala Ser Thr Thr Ala Pro Lys Val Tyr Pro Leu Ser Ser Cys Cys Gly
1 5 10 15
Asp Lys Ser Ser Ser Gly Val Thr Leu Gly Cys Leu Val Ser Ser Tyr
20 25 30
Met Pro Glu Pro Val Thr Val Thr Trp Asn Ser Gly Ala Leu Lys Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Met Val Thr Val Pro Ala Ser Ser Ser Gly Thr Gln Thr
65 70 75 80
Phe Thr Cys Asn Val Ala His Pro Ala Ser Ser Thr Lys Val Asp Lys
85 90 95
Ala Val Gly Val Ser Ser Asp Cys Ser Lys Pro Asn Asn Gln His Cys
100 105 110
Val Arg Glu Pro Ser Val Phe Ile Phe Pro Pro Lys Pro Lys Asp Thr
115 120 125
Leu Met Ile Thr Gly Thr Pro Glu Val Thr Cys Val Val Val Asn Val
130 135 140
Gly His Asp Asn Pro Glu Val Gln Phe Ser Trp Phe Val Asp Asp Val
145 150 155 160
Glu Val His Thr Ala Arg Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser
165 170 175
Thr Tyr Arg Val Val Ser Ala Leu Pro Ile Gln His Gln Asp Trp Thr
180 185 190
Gly Gly Lys Glu Phe Lys Cys Lys Val Asn Ile Lys Gly Leu Ser Ala
195 200 205
Ser Ile Val Arg Ile Ile Ser Arg Ser Lys Gly Pro Ala Arg Glu Pro
210 215 220
Gln Val Tyr Val Leu Asp Pro Pro Lys Glu Glu Leu Ser Lys Ser Thr
225 230 235 240
Val Ser Leu Thr Cys Met Val Ile Gly Phe Tyr Pro Glu Asp Val Asp
245 250 255
Val Glu Trp Gln Arg Asp Arg Gln Thr Glu Ser Glu Asp Lys Tyr Arg
260 265 270
Thr Thr Pro Pro Gln Leu Asp Ala Asp Arg Ser Tyr Phe Leu Tyr Ser
275 280 285
Lys Leu Arg Val Asp Arg Asn Ser Trp Gln Arg Gly Asp Thr Tyr Thr
290 295 300
Cys Val Val Met His Glu Ala Leu His Asn His Tyr Met Gln Lys Ser
305 310 315 320
Thr Ser Lys Ser Ala Gly Lys
325
<210> 77
<211> 984
<212> ДНК
<213> Bubalus arnee
<400> 77
gcctccacca cagccccgaa agtctaccct ctgagttctt gctgcgggga caagtccagc 60
tcgggggtga ccctgggctg cctggtctcc agctacatgc ccgagccggt gaccgtgacc 120
tggaactcgg gtgccctgaa gagcggcgtg cacaccttcc cggccgtcct tcagtcctcc 180
gggctctact ctctcagcag catggtgacc gtgcccgcca gcagctcagg aacccagacc 240
ttcacctgca acgtagccca cccggccagc agcaccaagg tggacaaggc tgttggggtc 300
tccagtgact gctccaagcc taataaccag cattgcgtga gggaaccatc tgtcttcatc 360
ttcccaccga aacccaaaga caccctgatg atcacaggaa cgcccgaggt cacgtgtgtg 420
gtggtgaacg tgggccacga taaccccgag gtgcagttct cctggttcgt ggacgacgtg 480
gaggtgcaca cggccaggac gaagccgaga gaggagcagt tcaacagcac gtaccgcgtg 540
gtcagcgccc tgcccatcca gcaccaggac tggactggag gaaaggagtt caagtgcaag 600
gtcaacatca aaggcctctc ggcctccatc gtgaggatca tctccaggag caaagggccg 660
gcccgggagc cgcaggtgta tgtcctggac ccacccaagg aagagctcag caaaagcacg 720
gtcagcctca cctgcatggt catcggcttc tacccagaag atgtagacgt ggagtggcag 780
agagaccggc agactgagtc ggaggacaag taccgcacga ccccgcccca gctggacgcc 840
gaccgctcct acttcctgta cagcaagctc agggtggaca ggaacagctg gcagagagga 900
gacacctaca cgtgtgtggt gatgcacgag gccctgcaca atcactacat gcagaagtcc 960
acctctaagt ctgcgggtaa atga 984
<210> 78
<211> 352
<212> Белок
<213> Bubalus arnee
<400> 78
Ala Ser Thr Thr Ala Pro Lys Val Tyr Pro Leu Ala Ser Ser Cys Gly
1 5 10 15
Asp Thr Ser Ser Ser Thr Val Thr Leu Gly Cys Leu Val Ser Ser Tyr
20 25 30
Met Pro Glu Pro Val Thr Val Thr Trp Asn Ser Gly Ala Leu Lys Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Arg Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Met Val Thr Val Pro Ala Ser Ser Ser Glu Thr Gln Thr
65 70 75 80
Phe Thr Cys Asn Val Ala His Pro Ala Ser Ser Thr Lys Val Asp Lys
85 90 95
Ala Val Thr Ala Arg Arg Pro Val Pro Thr Thr Pro Lys Thr Thr Ile
100 105 110
Pro Pro Gly Lys Pro Thr Thr Pro Lys Ser Glu Val Glu Lys Thr Pro
115 120 125
Cys Gln Cys Ser Lys Cys Pro Glu Pro Leu Gly Gly Leu Ser Val Phe
130 135 140
Ile Phe Pro Pro Lys Pro Lys Asp Thr Leu Thr Ile Ser Gly Thr Pro
145 150 155 160
Glu Val Thr Cys Val Val Val Asp Val Gly Gln Asp Asp Pro Glu Val
165 170 175
Gln Phe Ser Trp Phe Val Asp Asp Val Glu Val His Thr Ala Arg Thr
180 185 190
Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Ala
195 200 205
Leu Arg Ile Gln His Gln Asp Trp Leu Gln Gly Lys Glu Phe Lys Cys
210 215 220
Lys Val Asn Asn Lys Gly Leu Pro Ala Pro Ile Val Arg Thr Ile Ser
225 230 235 240
Arg Thr Lys Gly Gln Ala Arg Glu Pro Gln Val Tyr Val Leu Ala Pro
245 250 255
Pro Arg Glu Glu Leu Ser Lys Ser Thr Leu Ser Leu Thr Cys Leu Ile
260 265 270
Thr Gly Phe Tyr Pro Glu Glu Ile Asp Val Glu Trp Gln Arg Asn Gly
275 280 285
Gln Pro Glu Ser Glu Asp Lys Tyr His Thr Thr Ala Pro Gln Leu Asp
290 295 300
Ala Asp Gly Ser Tyr Phe Leu Tyr Ser Lys Leu Arg Val Asn Lys Ser
305 310 315 320
Ser Trp Gln Glu Gly Asp His Tyr Thr Cys Ala Val Met His Glu Ala
325 330 335
Leu Arg Asn His Tyr Lys Glu Lys Ser Ile Ser Arg Ser Pro Gly Lys
340 345 350
<210> 79
<211> 1059
<212> ДНК
<213> Bubalus arnee
<400> 79
gcctccacca cagccccgaa agtctaccct ctggcatcca gctgcggaga cacatccagc 60
tccaccgtga ccctgggctg cctggtctcc agctacatgc ccgagccggt gaccgtgacc 120
tggaactcgg gtgccctgaa gagcggcgtg cacaccttcc cggccgtccg gcagtcctct 180
gggctgtact ctctcagcag catggtgact gtgcccgcca gcagctcaga aacccagacc 240
ttcacctgca acgtagccca cccggccagc agcaccaagg tggacaaggc tgtcactgca 300
aggcgtccag tcccgacgac gccaaagaca actatccctc ctggaaaacc cacaacccca 360
aagtctgaag ttgaaaagac accctgccag tgttccaaat gcccagaacc tctgggagga 420
ctgtctgtct tcatcttccc accgaaaccc aaggacaccc tcacaatctc gggaacgccc 480
gaggtcacgt gtgtggtggt ggacgtgggc caggatgacc ccgaggtgca gttctcctgg 540
ttcgtggacg acgtggaggt gcacacggcc aggacgaagc cgagagagga gcagttcaac 600
agcacctacc gcgtggtcag cgccctgcgc atccagcacc aggactggct gcagggaaag 660
gagttcaagt gcaaggtcaa caacaaaggc ctcccggccc ccattgtgag gaccatctcc 720
aggaccaaag ggcaggcccg ggagccgcag gtgtatgtcc tggccccacc ccgggaagag 780
ctcagcaaaa gcacgctcag cctcacctgc ctgatcaccg gtttctaccc agaagagata 840
gacgtggagt ggcagagaaa tgggcagcct gagtcggagg acaagtacca cacgaccgca 900
ccccagctgg atgctgacgg ctcctacttc ctgtacagca agctcagggt gaacaagagc 960
agctggcagg aaggagacca ctacacgtgt gcagtgatgc acgaagcttt acggaatcac 1020
tacaaagaga agtccatctc gaggtctccg ggtaaatga 1059
<210> 80
<211> 352
<212> Белок
<213> Bubalus arnee
<400> 80
Ala Ser Thr Thr Ala Pro Lys Val Tyr Pro Leu Ala Ser Arg Cys Gly
1 5 10 15
Asp Thr Ser Ser Ser Thr Val Thr Leu Gly Cys Leu Val Ser Ser Tyr
20 25 30
Met Pro Glu Pro Val Thr Val Thr Trp Asn Ser Gly Ala Leu Lys Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Met Val Thr Val Pro Ala Ser Thr Ser Glu Thr Gln Thr
65 70 75 80
Phe Thr Cys Asn Val Ala His Pro Ala Ser Ser Thr Lys Val Asp Lys
85 90 95
Ala Val Thr Ala Arg Arg Pro Val Pro Thr Thr Pro Lys Thr Thr Ile
100 105 110
Pro Pro Gly Lys Pro Thr Thr Gln Glu Ser Glu Val Glu Lys Thr Pro
115 120 125
Cys Gln Cys Ser Lys Cys Pro Glu Pro Leu Gly Gly Leu Ser Val Phe
130 135 140
Ile Phe Pro Pro Lys Pro Lys Asp Thr Leu Thr Ile Ser Gly Thr Pro
145 150 155 160
Glu Val Thr Cys Val Val Val Asp Val Gly Gln Asp Asp Pro Glu Val
165 170 175
Gln Phe Ser Trp Phe Val Asp Asp Val Glu Val His Thr Ala Arg Thr
180 185 190
Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Ala
195 200 205
Leu Arg Ile Gln His Gln Asp Trp Leu Gln Gly Lys Glu Phe Lys Cys
210 215 220
Lys Val Asn Asn Lys Gly Leu Pro Ala Pro Ile Val Arg Thr Ile Ser
225 230 235 240
Arg Thr Lys Gly Gln Ala Arg Glu Pro Gln Val Tyr Val Leu Ala Pro
245 250 255
Pro Arg Glu Glu Leu Ser Lys Ser Thr Leu Ser Leu Thr Cys Leu Ile
260 265 270
Thr Gly Phe Tyr Pro Glu Glu Ile Asp Val Glu Trp Gln Arg Asn Gly
275 280 285
Gln Pro Glu Ser Glu Asp Lys Tyr His Thr Thr Ala Pro Gln Leu Asp
290 295 300
Ala Asp Gly Ser Tyr Phe Leu Tyr Ser Arg Leu Arg Val Asn Lys Ser
305 310 315 320
Ser Trp Gln Glu Gly Asp His Tyr Thr Cys Ala Val Met His Glu Ala
325 330 335
Leu Arg Asn His Tyr Lys Glu Lys Ser Ile Ser Arg Ser Pro Gly Lys
340 345 350
<210> 81
<211> 1059
<212> ДНК
<213> Bubalus arnee
<400> 81
gcctccacca cagccccgaa agtctaccct ctggcatccc gctgcggaga cacatccagc 60
tccaccgtga ccctgggctg cctggtctcc agctacatgc ccgagccggt gaccgtgacc 120
tggaactcgg gtgccctgaa gagtggcgtg cacaccttcc cggccgtcct tcagtcctcc 180
gggctgtact ctctcagcag catggtgacc gtgcccgcca gcacctcaga aacccagacc 240
ttcacctgca acgtagccca cccggccagc agcaccaagg tggacaaggc tgtcactgca 300
aggcgtccag tcccgacgac gccaaagaca accatccctc ctggaaaacc cacaacccag 360
gagtctgaag ttgaaaagac accctgccag tgttccaaat gcccagaacc tctgggagga 420
ctgtctgtct tcatcttccc accgaaaccc aaggacaccc tcacaatctc gggaacgccc 480
gaggtcacgt gtgtggtggt ggacgtgggc caggatgacc ccgaggtgca gttctcctgg 540
ttcgtggacg acgtggaggt gcacacggcc aggacgaagc cgagagagga gcagttcaac 600
agcacctacc gcgtggtcag cgccctgcgc atccagcacc aggactggct gcagggaaag 660
gagttcaagt gcaaggtcaa caacaaaggc ctcccggccc ccattgtgag gaccatctcc 720
aggaccaaag ggcaggcccg ggagccgcag gtgtatgtcc tggccccacc ccgggaagag 780
ctcagcaaaa gcacgctcag cctcacctgc ctgatcaccg gtttctaccc agaagagata 840
gacgtggagt ggcagagaaa tgggcagcct gagtcggagg acaagtacca cacgaccgca 900
ccccagctgg atgctgacgg ctcctacttc ctgtacagca ggctcagggt gaacaagagc 960
agctggcagg aaggagacca ctacacgtgt gcagtgatgc atgaagcttt acggaatcac 1020
tacaaagaga agtccatctc gaggtctccg ggtaaatga 1059
<210> 82
<211> 105
<212> Белок
<213> Bubalus arnee
<400> 82
Gln Pro Lys Ala Ser Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu
1 5 10 15
Glu Leu Gly Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe
20 25 30
Tyr Pro Ser Gly Val Thr Val Ala Trp Lys Ala Ser Gly Ser Pro Val
35 40 45
Thr Gln Gly Val Glu Thr Thr Lys Pro Ser Lys Gln Ser Asn Asn Lys
50 55 60
Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Asp Lys Trp Lys Ser
65 70 75 80
His Ser Ser Phe Ser Cys Leu Val Thr His Glu Gly Ser Thr Val Glu
85 90 95
Lys Lys Val Ala Pro Ala Glu Cys Ser
100 105
<210> 83
<211> 318
<212> ДНК
<213> Bubalus arnee
<400> 83
cagcccaagg cctccccctc ggtcacactc ttcccgccct cctctgagga gctcggcgcc 60
aacaaggcca ccctggtgtg cctcatcagc gacttctacc ccagcggcgt gacggtggcc 120
tggaaggcaa gcggcagccc cgtcacccag ggcgtggaga ccaccaagcc ctccaagcag 180
agcaacaaca agtacgcggc cagcagctac ctgagcctga cgcctgacaa gtggaaatct 240
cacagcagct tcagctgcct ggtcacgcac gaggggagca ccgtggagaa gaaggtggcc 300
cccgcagagt gctcttag 318
<210> 84
<211> 331
<212> Белок
<213> Bubalus arnee
<400> 84
Ala Ser Thr Thr Ala Pro Ser Val Phe Pro Leu Ala Pro Ser Cys Gly
1 5 10 15
Ser Thr Ser Gly Ser Thr Val Ala Leu Ala Cys Leu Val Ser Gly Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ser Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ser Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Thr Val Thr Val Pro Ser Ser Arg Trp Pro Ser Glu Thr
65 70 75 80
Phe Thr Cys Asn Val Val His Pro Ala Ser Asn Thr Lys Val Asp Lys
85 90 95
Pro Val Pro Lys Glu Ser Thr Cys Lys Cys Ile Ser Pro Cys Pro Val
100 105 110
Pro Glu Ser Leu Gly Gly Pro Ser Val Phe Ile Phe Pro Pro Lys Pro
115 120 125
Lys Asp Ile Leu Arg Ile Thr Arg Thr Pro Glu Ile Thr Cys Val Val
130 135 140
Leu Asp Leu Gly Arg Glu Asp Pro Glu Val Gln Ile Ser Trp Phe Val
145 150 155 160
Asp Gly Lys Glu Val His Thr Ala Lys Thr Gln Pro Arg Glu Gln Gln
165 170 175
Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu Pro Ile Glu His Gln
180 185 190
Asp Trp Leu Thr Gly Lys Glu Phe Lys Cys Arg Val Asn His Ile Gly
195 200 205
Leu Pro Ser Pro Ile Glu Arg Thr Ile Ser Lys Ala Arg Gly Gln Ala
210 215 220
His Gln Pro Ser Val Tyr Val Leu Pro Pro Ser Pro Lys Glu Leu Ser
225 230 235 240
Ser Ser Asp Thr Val Thr Leu Thr Cys Leu Ile Lys Asp Phe Phe Pro
245 250 255
Pro Glu Ile Asp Val Glu Trp Gln Ser Asn Gly Gln Pro Glu Pro Glu
260 265 270
Ser Lys Tyr His Thr Thr Ala Pro Gln Leu Asp Glu Asp Gly Ser Tyr
275 280 285
Phe Leu Tyr Ser Lys Leu Ser Val Asp Lys Ser Arg Trp Gln Gln Gly
290 295 300
Asp Thr Phe Thr Cys Ala Val Met His Glu Ala Leu Gln Asn His Tyr
305 310 315 320
Thr Asp Leu Ser Leu Ser His Ser Pro Gly Lys
325 330
<210> 85
<211> 996
<212> ДНК
<213> Bubalus arnee
<400> 85
gcctccacca cggccccctc ggttttccca ctggccccca gctgcgggtc cacttccggc 60
tccacggtgg ccctggcctg cctggtgtca ggctacttcc ccgagcctgt aactgtgtcc 120
tggaattccg gctccttgac cagcggtgtg cacaccttcc cgtccgtcct gcagtcctca 180
gggctctact ccctcagcag cacggtgaca gtgccctcca gcaggtggcc cagcgagacc 240
ttcacctgca acgtggtcca cccggccagc aacactaaag tagacaagcc agtgcccaaa 300
gagtccacct gcaagtgtat atccccatgc ccagtccctg aatcactggg agggccttcg 360
gtcttcatct ttcccccgaa acccaaggac atcctcagga ttacccgaac acccgagatc 420
acctgtgtgg tgttagatct gggccgtgag gaccctgagg tgcagatcag ctggttcgtg 480
gatggtaagg aggtgcacac agccaagacg cagcctcgtg agcagcagtt caacagcacc 540
taccgtgtgg tcagcgtcct ccccattgag caccaggact ggctcaccgg aaaggagttc 600
aagtgcagag tcaaccacat aggcctcccg tcccccatcg agaggactat ctccaaagcc 660
agagggcaag cccatcagcc cagtgtgtat gtcctgccac catccccaaa ggagttgtca 720
tccagtgaca cggtcaccct gacctgcctg atcaaagact tcttcccacc tgagattgat 780
gtggagtggc agagcaatgg acagccggag cccgagagca agtaccacac gactgcgccc 840
cagctggacg aggacgggtc ctacttcctg tacagcaagc tctctgtgga caagagccgc 900
tggcagcagg gagacacctt cacatgtgcg gtgatgcatg aagctctaca gaaccactac 960
acagatctat ccctctccca ttctccgggt aaatga 996
<210> 86
<211> 102
<212> Белок
<213> Bubalus arnee
<400> 86
Pro Ser Val Phe Leu Phe Lys Pro Ser Glu Glu Gln Leu Arg Thr Gly
1 5 10 15
Thr Val Ser Val Val Cys Leu Val Asn Asp Phe Tyr Pro Lys Asp Ile
20 25 30
Asn Val Lys Val Lys Val Asp Gly Val Thr Gln Asn Ser Asn Phe Gln
35 40 45
Asn Ser Phe Thr Asp Gln Asp Ser Lys Lys Ser Thr Tyr Ser Leu Ser
50 55 60
Ser Thr Leu Thr Leu Ser Ser Ser Glu Tyr Gln Ser His Asn Ala Tyr
65 70 75 80
Ala Cys Glu Val Ser His Lys Ser Leu Pro Thr Ala Leu Val Lys Ser
85 90 95
Phe Asn Lys Asn Glu Cys
100
<210> 87
<211> 309
<212> ДНК
<213> Bubalus arnee
<400> 87
ccatccgtct tcctcttcaa accatctgag gaacagctga ggaccggaac tgtctctgtc 60
gtgtgcttgg tgaatgattt ctaccccaaa gatatcaatg tcaaggtgaa agtggatggg 120
gttacccaga acagcaactt ccagaacagc ttcacagacc aggacagcaa gaaaagcacc 180
tacagcctca gcagcaccct gacactgtcc agctcagagt accagagcca taacgcctat 240
gcgtgtgagg tcagccacaa gagcctgccc accgccctcg tcaagagctt caataagaat 300
gaatgttag 309
<210> 88
<211> 106
<212> Белок
<213> Bubalus arnee
<400> 88
Gly Gln Pro Lys Ser Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Thr
1 5 10 15
Glu Glu Leu Ser Thr Asn Lys Ala Thr Val Val Cys Leu Ile Asn Asp
20 25 30
Phe Tyr Pro Gly Ser Val Asn Val Val Trp Lys Ala Asp Gly Ser Thr
35 40 45
Ile Asn Gln Asn Val Lys Thr Thr Gln Ala Ser Lys Gln Ser Asn Ser
50 55 60
Lys Tyr Ala Ala Ser Ser Tyr Leu Thr Leu Thr Gly Ser Glu Trp Lys
65 70 75 80
Ser Lys Ser Ser Tyr Thr Cys Glu Val Thr His Glu Gly Ser Thr Val
85 90 95
Thr Lys Thr Val Lys Pro Ser Glu Cys Ser
100 105
<210> 89
<211> 321
<212> ДНК
<213> Bubalus arnee
<400> 89
ggtcagccca agtccgcacc ctcggtcacc ctgttcccgc cttccacgga ggagctcagt 60
accaacaagg ccaccgtggt gtgtctcatc aacgacttct acccgggtag cgtgaacgtg 120
gtctggaagg cagatggcag caccatcaat cagaacgtga agaccaccca ggcctccaaa 180
cagagcaaca gcaagtacgc ggccagcagc tacctgaccc tgacgggcag cgagtggaag 240
tctaagagca gttacacctg cgaggtcacg cacgagggga gcaccgtgac gaagacagtg 300
aagccctcag agtgttctta g 321
<210> 90
<211> 331
<212> Белок
<213> Bubalus arnee
<400> 90
Ala Ser Thr Thr Pro Pro Lys Val Tyr Pro Leu Thr Ser Cys Cys Gly
1 5 10 15
Asp Thr Ser Ser Ser Ile Val Thr Leu Gly Cys Leu Val Ser Ser Tyr
20 25 30
Met Pro Glu Pro Val Thr Val Thr Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Ile Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ala Ser Thr Ser Gly Ala Gln Thr
65 70 75 80
Phe Ile Cys Asn Val Ala His Pro Ala Ser Ser Thr Lys Val Asp Lys
85 90 95
Arg Val Glu Pro Gly Cys Pro Asp Pro Cys Lys His Cys Arg Cys Pro
100 105 110
Pro Pro Glu Leu Pro Gly Gly Pro Ser Val Phe Ile Phe Pro Pro Lys
115 120 125
Pro Lys Asp Thr Leu Thr Ile Ser Gly Thr Pro Glu Val Thr Cys Val
130 135 140
Val Val Asp Val Gly Gln Asp Asp Pro Glu Val Gln Phe Ser Trp Phe
145 150 155 160
Val Asp Asn Val Glu Val Arg Thr Ala Arg Thr Lys Pro Arg Glu Glu
165 170 175
Gln Phe Asn Ser Thr Phe Arg Val Val Ser Ala Leu Pro Ile Gln His
180 185 190
Gln Asp Trp Thr Gly Gly Lys Glu Phe Lys Cys Lys Val His Asn Glu
195 200 205
Ala Leu Pro Ala Pro Ile Val Arg Thr Ile Ser Arg Thr Lys Gly Gln
210 215 220
Ala Arg Glu Pro Gln Val Tyr Val Leu Ala Pro Pro Gln Glu Glu Leu
225 230 235 240
Ser Lys Ser Thr Leu Ser Val Thr Cys Leu Val Thr Gly Phe Tyr Pro
245 250 255
Asp Tyr Ile Ala Val Glu Trp Gln Lys Asn Gly Gln Pro Glu Ser Glu
260 265 270
Asp Lys Tyr Gly Thr Thr Thr Ser Gln Leu Asp Ala Asp Gly Ser Tyr
275 280 285
Phe Leu Tyr Ser Arg Leu Arg Val Asp Lys Asn Ser Trp Gln Glu Gly
290 295 300
Asp Thr Tyr Ala Cys Val Val Met His Glu Ala Leu His Asn His Tyr
305 310 315 320
Thr Gln Lys Ser Ile Ser Lys Pro Pro Gly Lys
325 330
<210> 91
<211> 996
<212> ДНК
<213> Bubalus arnee
<400> 91
gcctcaacaa cacccccgaa agtctaccct ctgacttctt gctgcgggga cacgtccagc 60
tccatcgtga ccctgggctg cctggtctcc agctatatgc ccgagccggt gaccgtgacc 120
tggaactctg gtgccctgac cagcggcgtg cacaccttcc cggccatcct gcagtcctcc 180
gggctctact ctctcagcag cgtggtgacc gtgccggcca gcacctcagg agcccagacc 240
ttcatctgca acgtagccca cccggccagc agcaccaagg tggacaagcg tgttgagccc 300
ggatgcccgg acccatgcaa acattgccga tgcccacccc ctgagctccc cggaggaccg 360
tctgtcttca tcttcccacc gaaacccaag gacaccctta caatctctgg aacgcccgag 420
gtcacgtgtg tggtggtgga cgtgggccag gatgaccccg aggtgcagtt ctcctggttc 480
gtggacaacg tggaggtgcg cacggccagg acaaagccga gagaggagca gttcaacagc 540
accttccgcg tggtcagcgc cctgcccatc cagcaccaag actggactgg aggaaaggag 600
ttcaagtgca aggtccacaa cgaagccctc ccggccccca tcgtgaggac catctccagg 660
accaaagggc aggcccggga gccgcaggtg tacgtcctgg ccccacccca ggaagagctc 720
agcaaaagca cgctcagcgt cacctgcctg gtcaccggct tctacccaga ctacatcgcc 780
gtggagtggc agaaaaatgg gcagcctgag tcggaggaca agtacggcac gaccacatcc 840
cagctggacg ccgacggctc ctacttcctg tacagcaggc tcagggtgga caagaacagc 900
tggcaagaag gagacaccta cgcgtgtgtg gtgatgcacg aggctctgca caaccactac 960
acacagaagt cgatctctaa gcctccgggt aaatga 996
<210> 92
<211> 329
<212> Белок
<213> Bubalus arnee
<400> 92
Ala Ser Thr Thr Ala Pro Lys Val Tyr Pro Leu Thr Ser Cys Cys Gly
1 5 10 15
Asp Thr Ser Ser Ser Ser Ser Ile Val Thr Leu Gly Cys Leu Val Ser
20 25 30
Ser Tyr Met Pro Glu Pro Val Thr Val Thr Trp Asn Ser Gly Ala Leu
35 40 45
Thr Ser Gly Val His Thr Phe Pro Ala Ile Leu Gln Ser Ser Gly Leu
50 55 60
Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ala Ser Thr Ser Gly Ala
65 70 75 80
Gln Thr Phe Ile Cys Asn Val Ala His Pro Ala Ser Ser Ala Lys Val
85 90 95
Asp Lys Arg Val Gly Ile Ser Ser Asp Tyr Ser Lys Cys Ser Lys Pro
100 105 110
Pro Cys Val Ser Arg Pro Ser Val Phe Ile Phe Pro Pro Lys Pro Lys
115 120 125
Asp Ser Leu Met Ile Thr Gly Thr Pro Glu Val Thr Cys Val Val Val
130 135 140
Asp Val Gly Gln Gly Asp Pro Glu Val Gln Phe Ser Trp Phe Val Asp
145 150 155 160
Asn Val Glu Val Arg Thr Ala Arg Thr Lys Pro Arg Glu Glu Gln Phe
165 170 175
Asn Ser Thr Phe Arg Val Val Ser Ala Leu Pro Ile Gln His Asp His
180 185 190
Trp Thr Gly Gly Lys Glu Phe Lys Cys Lys Val His Ser Lys Gly Leu
195 200 205
Pro Ala Pro Ile Val Arg Thr Ile Ser Arg Ala Lys Gly Gln Ala Arg
210 215 220
Glu Pro Gln Val Tyr Val Leu Ala Pro Pro Gln Glu Glu Leu Ser Lys
225 230 235 240
Ser Thr Leu Ser Val Thr Cys Leu Val Thr Gly Phe Tyr Pro Asp Tyr
245 250 255
Ile Ala Val Glu Trp Gln Arg Ala Arg Gln Pro Glu Ser Glu Asp Lys
260 265 270
Tyr Gly Thr Thr Thr Ser Gln Leu Asp Ala Asp Gly Ser Tyr Phe Leu
275 280 285
Tyr Ser Arg Leu Arg Val Asp Lys Ser Ser Trp Gln Arg Gly Asp Thr
290 295 300
Tyr Ala Cys Val Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
305 310 315 320
Lys Ser Ile Ser Lys Pro Pro Gly Lys
325
<210> 93
<211> 990
<212> ДНК
<213> Bubalus arnee
<400> 93
gcctccacca cagccccgaa agtctaccct ctgacttctt gctgcgggga cacgtccagc 60
tccagctcca tcgtgaccct gggctgcctg gtctccagct atatgcccga gccggtgacc 120
gtgacctgga actctggtgc cctgaccagc ggcgtgcaca ccttcccggc catcctgcag 180
tcctccgggc tctactctct cagcagcgtg gtgaccgtgc cggccagcac ctcaggagcc 240
cagaccttca tctgcaacgt agcccacccg gccagcagcg ccaaggtgga caagcgtgtt 300
gggatctcca gtgactactc caagtgttct aaaccgcctt gcgtgagccg accgtctgtc 360
ttcatcttcc ccccgaaacc caaggacagc ctcatgatca caggaacgcc cgaggtcacg 420
tgtgtggtgg tggacgtggg ccagggtgac cccgaggtgc agttctcctg gttcgtggac 480
aacgtggagg tgcgcacggc caggacaaag ccgagagagg agcagttcaa cagcaccttc 540
cgcgtggtca gcgccctgcc catccagcac gaccactgga ctggaggaaa ggagttcaag 600
tgcaaggtcc acagcaaagg cctcccggcc cccatcgtga ggaccatctc cagggccaaa 660
gggcaggccc gggagccgca ggtgtacgtc ctggccccac cccaggaaga gctcagcaaa 720
agcacgctca gcgtcacctg cctggtcacc ggcttctacc cagactacat cgccgtggag 780
tggcagagag cgcggcagcc tgagtcggag gacaagtacg gcacgaccac atcccagctg 840
gacgccgacg gctcctactt cctgtacagc aggctcaggg tggacaagag cagctggcaa 900
agaggagaca cctacgcgtg tgtggtgatg cacgaggctc tgcacaacca ctacacacag 960
aagtcgatct ctaagcctcc gggtaaatga 990
<210> 94
<211> 328
<212> Белок
<213> Bubalus arnee
<400> 94
Ala Pro Lys Thr Ala Pro Ser Val Tyr Pro Leu Ala Pro Cys Gly Arg
1 5 10 15
Asp Thr Ser Gly Pro Asn Val Ala Leu Gly Cys Leu Ala Ser Ser Tyr
20 25 30
Phe Pro Glu Pro Val Thr Met Thr Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ser Val Leu Gln Pro Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Met Val Thr Val Pro Ala Ser Ser Leu Ser Ser Lys Ser
65 70 75 80
Tyr Thr Cys Asn Val Asn His Pro Ala Thr Thr Thr Lys Val Asp Lys
85 90 95
Arg Val Gly Thr Lys Thr Lys Pro Pro Cys Pro Ile Cys Pro Gly Cys
100 105 110
Glu Val Ala Gly Pro Ser Val Phe Ile Phe Pro Pro Lys Pro Lys Asp
115 120 125
Thr Leu Met Ile Ser Gln Thr Pro Glu Val Thr Cys Val Val Val Asp
130 135 140
Val Ser Lys Glu His Ala Glu Val Gln Phe Ser Trp Tyr Val Asp Gly
145 150 155 160
Val Glu Val His Thr Ala Glu Thr Arg Pro Lys Glu Glu Gln Phe Asn
165 170 175
Ser Thr Tyr Arg Val Val Ser Val Leu Pro Ile Gln His Gln Asp Trp
180 185 190
Leu Lys Gly Lys Glu Phe Lys Cys Lys Val Asn Asn Val Asp Leu Pro
195 200 205
Ala Pro Ile Thr Arg Thr Ile Ser Lys Ala Ile Gly Gln Ser Arg Glu
210 215 220
Pro Gln Val Tyr Thr Leu Pro Pro Pro Ala Glu Glu Leu Ser Arg Ser
225 230 235 240
Lys Val Thr Val Thr Cys Leu Val Ile Gly Phe Tyr Pro Pro Asp Ile
245 250 255
His Val Glu Trp Lys Ser Asn Gly Gln Pro Glu Pro Glu Gly Asn Tyr
260 265 270
Arg Thr Thr Pro Pro Gln Gln Asp Val Asp Gly Thr Phe Phe Leu Tyr
275 280 285
Ser Lys Leu Ala Val Asp Lys Ala Arg Trp Asp His Gly Glu Thr Phe
290 295 300
Glu Cys Ala Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
305 310 315 320
Ser Ile Ser Lys Thr Gln Gly Lys
325
<210> 95
<211> 987
<212> ДНК
<213> Bubalus arnee
<400> 95
gcccccaaga cggccccatc ggtctaccct ctggccccct gcggcaggga cacgtctggc 60
cctaacgtgg ccttgggctg cctggcctca agctacttcc ccgagccagt gaccatgacc 120
tggaactcgg gcgccctgac cagtggcgtg cataccttcc catccgtcct gcagccgtca 180
gggctctact ccctcagcag catggtgacc gtgccggcca gcagcctgtc cagcaagagc 240
tacacctgca atgtcaacca cccggccacc accaccaagg tggacaagcg tgttggaaca 300
aagaccaaac caccatgtcc catatgccca ggctgtgaag tggccgggcc ctcggtcttc 360
atcttccctc caaaacccaa ggacaccctc atgatctccc agacccccga ggtcacgtgc 420
gtggtggtgg acgtcagcaa ggagcacgcc gaggtccagt tctcctggta cgtggacggc 480
gtagaggtgc acacggccga gacgagacca aaggaggagc agttcaacag cacctaccgt 540
gtggtcagcg tcctgcccat ccagcaccag gactggctga aggggaagga gttcaagtgc 600
aaggtcaaca acgtagacct cccagccccc atcacgagga ccatctccaa ggctataggg 660
cagagccggg agccgcaggt gtacaccctg cccccacccg ccgaggagct gtccaggagc 720
aaagtcaccg taacctgcct ggtcattggc ttctacccac ctgacatcca tgttgagtgg 780
aagagcaacg gacagccgga gccagagggc aattaccgca ccaccccgcc ccagcaggac 840
gtggacggga ccttcttcct gtacagcaag ctcgcggtgg acaaggcaag atgggaccat 900
ggagaaacat ttgagtgtgc ggtgatgcac gaggctctgc acaaccacta cacccagaag 960
tccatctcca agactcaggg taaatga 987
<210> 96
<211> 328
<212> Белок
<213> Bubalus arnee
<400> 96
Ala Pro Lys Thr Ala Pro Ser Val Tyr Pro Leu Ala Pro Cys Gly Arg
1 5 10 15
Asp Val Ser Gly Pro Asn Val Ala Leu Gly Cys Leu Ala Ser Ser Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Thr Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ser Val Leu Gln Pro Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Met Val Thr Val Pro Ala Ser Ser Leu Ser Ser Lys Ser
65 70 75 80
Tyr Thr Cys Asn Val Asn His Pro Ala Thr Thr Thr Lys Val Asp Lys
85 90 95
Arg Val Gly Ile His Gln Pro Gln Thr Cys Pro Ile Cys Pro Gly Cys
100 105 110
Glu Val Ala Gly Pro Ser Val Phe Ile Phe Pro Pro Lys Pro Lys Asp
115 120 125
Thr Leu Met Ile Ser Gln Thr Pro Glu Val Thr Cys Val Val Val Asp
130 135 140
Val Ser Lys Glu His Ala Glu Val Gln Phe Ser Trp Tyr Val Asp Gly
145 150 155 160
Val Glu Val His Thr Ala Glu Thr Arg Pro Lys Glu Glu Gln Phe Asn
165 170 175
Ser Thr Tyr Arg Val Val Ser Val Leu Pro Ile Gln His Gln Asp Trp
180 185 190
Leu Lys Gly Lys Glu Phe Lys Cys Lys Val Asn Asn Val Asp Leu Pro
195 200 205
Ala Pro Ile Thr Arg Thr Ile Ser Lys Ala Ile Gly Gln Ser Arg Glu
210 215 220
Pro Gln Val Tyr Thr Leu Pro Pro Pro Ala Glu Glu Leu Ser Arg Ser
225 230 235 240
Lys Val Thr Leu Thr Cys Leu Val Ile Gly Phe Tyr Pro Pro Asp Ile
245 250 255
His Val Glu Trp Lys Ser Asn Gly Gln Pro Glu Pro Glu Asn Thr Tyr
260 265 270
Arg Thr Thr Pro Pro Gln Gln Asp Val Asp Gly Thr Phe Phe Leu Tyr
275 280 285
Ser Lys Leu Ala Val Asp Lys Ala Arg Trp Asp His Gly Asp Lys Phe
290 295 300
Glu Cys Ala Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
305 310 315 320
Ser Ile Ser Lys Thr Gln Gly Lys
325
<210> 97
<211> 987
<212> ДНК
<213> Bubalus arnee
<400> 97
gcccccaaga cggccccatc ggtctaccct ctggccccct gcggcaggga cgtgtctggc 60
cctaacgtgg ccttgggctg cctggcctca agctacttcc ccgagccagt gaccgtgacc 120
tggaactcgg gcgccctgac cagtggcgtg cacaccttcc catccgtcct gcagccgtca 180
gggctctact ccctcagcag catggtgacc gtgccggcca gcagcctgtc cagcaagagc 240
tacacctgca atgtcaacca cccggccacc accaccaagg tggacaagcg tgttggaata 300
caccagccgc aaacatgtcc catatgccca ggctgtgaag tggccgggcc ctcggtcttc 360
atcttccctc caaaacccaa ggacaccctc atgatctccc agacccccga ggtcacgtgc 420
gtggtggtgg acgtcagcaa ggagcacgcc gaggtccagt tctcctggta cgtggacggc 480
gtagaggtgc acacggccga gacgagacca aaggaggagc agttcaacag cacctaccgt 540
gtggtcagcg tcctgcccat ccagcaccag gactggctga aggggaagga gttcaagtgc 600
aaggtcaaca acgtagacct cccagccccc atcacgagga ccatctccaa ggctataggg 660
cagagccggg agccgcaggt gtacaccctg cccccacccg ccgaggagct gtccaggagc 720
aaagtcacgc taacctgcct ggtcattggc ttctacccac ctgacatcca tgttgagtgg 780
aagagcaacg gacagccgga gccagagaac acataccgca ccaccccgcc ccagcaggac 840
gtggacggga ccttcttcct gtacagcaaa ctcgcggtgg acaaggcaag atgggaccat 900
ggagacaaat ttgagtgtgc ggtgatgcac gaggctctgc acaaccacta cacccagaag 960
tccatctcca agactcaggg taaatga 987
<210> 98
<211> 328
<212> Белок
<213> Bubalus arnee
<400> 98
Ala Pro Lys Thr Ala Pro Ser Val Tyr Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Asp Thr Ser Gly Pro Asn Val Ala Leu Gly Cys Leu Ala Ser Ser Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Thr Trp Asn Ser Gly Ala Leu Ser Ser
35 40 45
Gly Val His Thr Phe Pro Ser Val Leu Gln Pro Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Met Val Thr Val Pro Ala Ser Ser Leu Ser Ser Lys Ser
65 70 75 80
Tyr Thr Cys Asn Val Asn His Pro Ala Thr Thr Thr Lys Val Asp Lys
85 90 95
Arg Val Gly Thr Lys Thr Lys Pro Pro Cys Pro Ile Cys Pro Ala Cys
100 105 110
Glu Ser Pro Gly Pro Ser Val Phe Ile Phe Pro Pro Lys Pro Lys Asp
115 120 125
Thr Leu Met Ile Ser Arg Thr Pro Gln Val Thr Cys Val Val Val Asp
130 135 140
Val Ser Gln Glu Asn Pro Glu Val Gln Phe Ser Trp Tyr Val Asp Gly
145 150 155 160
Val Glu Val His Thr Ala Gln Thr Arg Pro Lys Glu Glu Gln Phe Asn
165 170 175
Ser Thr Tyr Arg Val Val Ser Val Leu Pro Ile Gln His Gln Asp Trp
180 185 190
Leu Asn Gly Lys Glu Phe Lys Cys Lys Val Asn Asn Lys Asp Leu Pro
195 200 205
Ala Pro Ile Thr Arg Ile Ile Ser Lys Ala Lys Gly Gln Thr Arg Glu
210 215 220
Pro Gln Val Tyr Thr Leu Pro Pro His Ala Glu Glu Leu Ser Arg Ser
225 230 235 240
Lys Val Ser Ile Thr Cys Leu Val Ile Gly Phe Tyr Pro Pro Asp Ile
245 250 255
Asp Val Glu Trp Gln Arg Asn Gly Gln Pro Glu Pro Glu Gly Asn Tyr
260 265 270
Arg Thr Thr Pro Pro Gln Gln Asp Val Asp Gly Thr Tyr Phe Leu Tyr
275 280 285
Ser Lys Phe Ser Val Asp Lys Ala Ser Trp Gln Gly Gly Gly Ile Phe
290 295 300
Gln Cys Ala Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
305 310 315 320
Ser Ile Ser Lys Thr Pro Gly Lys
325
<210> 99
<211> 987
<212> ДНК
<213> Bubalus arnee
<400> 99
gcccccaaga cggccccatc ggtctaccct ctggccccct gcagcaggga cacgtctggc 60
cctaacgtgg ccttgggctg cctggcctca agctacttcc ccgagccagt gaccgtgacc 120
tggaactcgg gcgccctgtc cagtggcgtg cataccttcc catccgtcct gcagccgtca 180
gggctctact ccctcagcag catggtgacc gtgccggcca gcagcctgtc cagcaagagc 240
tacacctgca atgtcaacca cccggccacc accaccaagg tggacaagcg tgttggaaca 300
aagaccaaac caccatgtcc catatgccca gcctgtgaat caccagggcc ctcggtcttc 360
atcttccctc caaaacccaa ggacaccctc atgatctccc ggacacccca ggtcacgtgc 420
gtggtggttg atgtgagcca ggagaacccg gaggtccagt tctcctggta cgtggacggc 480
gtagaggtgc acacggccca gacgaggcca aaggaggagc agttcaacag cacctaccgc 540
gtggtcagcg tcctacccat ccagcaccag gactggctga acgggaagga gttcaagtgc 600
aaggtcaaca acaaagacct cccagccccc atcacaagga tcatctccaa ggccaaaggg 660
cagacccggg agccgcaggt gtacaccctg cccccacacg ccgaggagct gtccaggagc 720
aaagtcagca taacctgcct ggtcattggc ttctacccac ctgacatcga tgtcgagtgg 780
caaagaaacg gacagccgga gccagagggc aattaccgca ccaccccgcc ccagcaggac 840
gtggacggga cctacttcct gtacagcaag ttctcggtgg acaaggccag ctggcagggt 900
ggaggcatat tccagtgtgc ggtgatgcac gaggctctgc acaaccacta cacccagaag 960
tctatctcca agactccggg taaatga 987
<210> 100
<211> 328
<212> Белок
<213> Bubalus arnee
<400> 100
Ala Pro Lys Thr Ala Pro Leu Val Tyr Pro Leu Ala Pro Cys Gly Arg
1 5 10 15
Asp Thr Ser Gly Pro Asn Val Ala Leu Gly Cys Leu Ala Ser Ser Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Thr Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ser Val Leu Gln Pro Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Met Val Thr Val Pro Ala Ser Ser Leu Ser Ser Lys Ser
65 70 75 80
Tyr Thr Cys Asn Val Asn His Pro Ala Thr Thr Thr Lys Val Asp Lys
85 90 95
Arg Val Gly Thr Lys Thr Lys Pro Pro Cys Pro Ile Cys Pro Ala Cys
100 105 110
Glu Ser Pro Gly Pro Ser Val Phe Ile Phe Pro Pro Lys Pro Lys Asp
115 120 125
Thr Leu Met Ile Ser Arg Thr Pro Gln Val Thr Cys Val Val Val Asp
130 135 140
Val Ser Gln Glu Asn Pro Glu Val Gln Phe Ser Trp Tyr Val Asp Gly
145 150 155 160
Val Glu Val His Thr Ala Gln Thr Arg Pro Lys Glu Glu Gln Phe Asn
165 170 175
Ser Thr Tyr Arg Val Val Ser Val Leu Pro Ile Gln His Gln Asp Trp
180 185 190
Leu Asn Gly Lys Glu Phe Lys Cys Lys Val Asn Asn Lys Asp Leu Pro
195 200 205
Ala Pro Ile Thr Arg Ile Ile Ser Lys Ala Lys Gly Gln Thr Arg Glu
210 215 220
Pro Gln Val Tyr Thr Leu Pro Pro His Ala Glu Glu Leu Ser Arg Ser
225 230 235 240
Lys Val Ser Ile Thr Cys Leu Val Ile Gly Phe Tyr Pro Pro Asp Ile
245 250 255
Asp Val Glu Trp Gln Arg Asn Gly Gln Pro Glu Pro Glu Gly Asn Tyr
260 265 270
Arg Thr Thr Pro Pro Gln Gln Asp Val Asp Gly Thr Tyr Phe Leu Tyr
275 280 285
Ser Lys Phe Ser Val Asp Lys Ala Ser Trp Gln Gly Gly Gly Ile Phe
290 295 300
Gln Cys Ala Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
305 310 315 320
Ser Ile Ser Lys Thr Pro Gly Lys
325
<210> 101
<211> 987
<212> ДНК
<213> Bubalus arnee
<400> 101
gcccccaaga cggccccatt ggtctaccct ctggccccct gcggcaggga cacgtctggc 60
cctaacgtgg ccttgggctg cctggcctca agctacttcc ccgagccagt gaccgtgacc 120
tggaactcgg gcgccctgac cagtggcgtg cataccttcc catccgtcct gcagccgtca 180
gggctctact ccctcagcag catggtgacc gtgccggcca gcagcctgtc cagcaagagc 240
tacacctgca atgtcaacca cccggccacc accaccaagg tggacaagcg tgttggaaca 300
aagaccaaac caccatgtcc catatgccca gcctgtgaat cgccagggcc ctcggtcttc 360
atcttccctc caaaacccaa ggacaccctc atgatctccc ggacacccca ggtcacgtgc 420
gtggtagttg atgtgagcca ggagaacccg gaggtccagt tctcctggta cgtggacggc 480
gtagaggtgc acacggccca gacgaggcca aaggaggagc agttcaacag cacctaccgc 540
gtggtcagcg tcctgcccat ccagcaccag gactggctga acgggaagga gttcaagtgc 600
aaggtcaaca acaaagacct cccagccccc atcacaagga tcatctccaa ggccaaaggg 660
cagacccggg agccgcaggt gtacaccctg cccccacacg ccgaggagct gtccaggagc 720
aaagtcagca taacctgcct ggtcattggc ttctacccac ctgacatcga tgtcgagtgg 780
caaagaaacg gacagccgga gccagagggc aattaccgca ccaccccgcc ccagcaggac 840
gtggacggga cctacttcct gtacagcaag ttctcggtgg acaaggccag ctggcagggt 900
ggaggcatat tccagtgtgc ggtgatgcac gaggctctgc acaaccacta cacccagaag 960
tctatctcca agactccggg taaatga 987
<210> 102
<211> 333
<212> Белок
<213> Bubalus arnee
<400> 102
Ala Tyr Asn Thr Ala Pro Ser Val Tyr Pro Leu Ala Pro Cys Gly Arg
1 5 10 15
Asp Val Ser Asp His Asn Val Ala Leu Gly Cys Leu Val Ser Ser Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Thr Trp Asn Ser Gly Ala Leu Ser Arg
35 40 45
Val Val His Thr Phe Pro Ser Val Leu Gln Pro Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Met Val Ile Val Ala Ala Ser Ser Leu Ser Thr Leu Ser
65 70 75 80
Tyr Thr Cys Asn Val Tyr His Pro Ala Thr Asn Thr Lys Val Asp Lys
85 90 95
Arg Val Asp Ile Glu Pro Pro Thr Pro Ile Cys Pro Glu Ile Cys Ser
100 105 110
Cys Pro Ala Ala Glu Val Leu Gly Ala Pro Ser Val Phe Leu Phe Pro
115 120 125
Pro Lys Pro Lys Asp Ile Leu Met Ile Ser Arg Thr Pro Lys Val Thr
130 135 140
Cys Val Val Val Asp Val Ser Gln Glu Glu Ala Glu Val Gln Phe Ser
145 150 155 160
Trp Tyr Val Asp Gly Val Gln Leu Tyr Thr Ala Gln Thr Arg Pro Met
165 170 175
Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu Pro Ile
180 185 190
Gln His Gln Asp Trp Leu Lys Gly Lys Glu Phe Lys Cys Lys Val Asn
195 200 205
Asn Lys Asp Leu Leu Ser Pro Ile Thr Arg Thr Ile Ser Lys Ala Thr
210 215 220
Gly Pro Ser Arg Val Pro Gln Val Tyr Thr Leu Pro Pro Ala Trp Glu
225 230 235 240
Glu Leu Ser Lys Ser Lys Val Ser Ile Thr Cys Leu Val Thr Gly Phe
245 250 255
Tyr Pro Pro Asp Ile Asp Val Glu Trp Gln Ser Asn Gly Gln Gln Glu
260 265 270
Pro Glu Gly Asn Tyr Arg Thr Thr Pro Pro Gln Gln Asp Val Asp Gly
275 280 285
Thr Tyr Phe Leu Tyr Ser Lys Leu Ala Val Asp Lys Val Arg Trp Gln
290 295 300
Arg Gly Asp Leu Phe Gln Cys Ala Val Met His Glu Ala Leu His Asn
305 310 315 320
His Tyr Thr Gln Lys Ser Ile Ser Lys Thr Gln Gly Lys
325 330
<210> 103
<211> 1002
<212> ДНК
<213> Bubalus arnee
<400> 103
gcctacaaca cagctccatc ggtctaccct ctggccccct gtggcaggga cgtgtctgat 60
cataacgtgg ccttgggctg ccttgtctca agctacttcc ccgagccagt gaccgtgacc 120
tggaactcgg gtgccctgtc cagagtcgtg cataccttcc catccgtcct gcagccgtca 180
gggctctact ccctcagcag catggtgatc gtggcggcca gcagcctgtc caccctgagc 240
tacacgtgca acgtctacca cccggccacc aacaccaagg tggacaagcg tgttgacatc 300
gaacccccca cacccatctg tcccgaaatt tgctcatgcc cagctgcaga ggtcctggga 360
gcaccgtcgg tcttcctctt ccctccaaaa cccaaggaca tcctcatgat ctcccggaca 420
cccaaggtca cgtgcgtggt ggtggacgtg agccaggagg aggctgaagt ccagttctcc 480
tggtacgtgg acggcgtaca gttgtacacg gcccagacga ggccaatgga ggagcagttc 540
aacagcacct accgcgtggt cagcgtcctg cccatccagc accaggactg gctgaagggg 600
aaggagttca agtgcaaggt caacaacaaa gacctccttt cccccatcac gaggaccatc 660
tccaaggcta cagggccgag ccgggtgccg caggtgtaca ccctgccccc agcctgggaa 720
gagctgtcca agagcaaagt cagcataacc tgcctggtca ctggcttcta cccacctgac 780
atcgatgtcg agtggcagag caacggacaa caagagccag agggcaatta ccgcaccacc 840
ccgccccagc aggacgtgga tgggacctac ttcctgtaca gcaagctcgc ggtggacaag 900
gtcaggtggc agcgtggaga cctattccag tgtgcggtga tgcacgaggc tctgcacaac 960
cactacaccc agaagtccat ctccaagact cagggtaaat ga 1002
<210> 104
<211> 277
<212> Белок
<213> Bubalus arnee
<400> 104
Thr Phe Pro Ser Val Leu Gln Pro Ser Gly Leu Tyr Ser Leu Ser Ser
1 5 10 15
Met Val Thr Val Pro Ala Ser Ser Leu Ser Ser Lys Ser Tyr Thr Cys
20 25 30
Asn Val Asn His Pro Ala Thr Thr Thr Lys Val Asp Lys Arg Val Gly
35 40 45
Thr Lys Thr Lys Pro Pro Cys Pro Ile Cys Pro Ala Cys Glu Gly Pro
50 55 60
Gly Pro Ser Ala Phe Ile Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
65 70 75 80
Ile Ser Arg Thr Pro Lys Val Thr Cys Val Val Val Asp Val Ser Gln
85 90 95
Glu Asn Pro Glu Val Gln Phe Ser Trp Tyr Val Asp Gly Val Glu Val
100 105 110
His Thr Ala Gln Thr Arg Pro Lys Glu Glu Gln Phe Asn Ser Thr Tyr
115 120 125
Arg Val Val Ser Val Leu Pro Ile Gln His Gln Asp Trp Leu Asn Gly
130 135 140
Lys Glu Phe Lys Cys Lys Val Asn Asn Lys Asp Leu Pro Ala Pro Ile
145 150 155 160
Thr Arg Ile Ile Ser Lys Ala Lys Gly Gln Thr Arg Glu Pro Gln Val
165 170 175
Tyr Thr Leu Pro Pro Pro Thr Glu Glu Leu Ser Arg Ser Lys Val Thr
180 185 190
Leu Thr Cys Leu Val Thr Gly Phe Tyr Pro Pro Asp Ile Asp Val Glu
195 200 205
Trp Gln Arg Asn Gly Gln Pro Glu Pro Glu Gly Asn Tyr Arg Thr Thr
210 215 220
Pro Pro Gln Gln Asp Val Asp Gly Thr Tyr Phe Leu Tyr Ser Lys Leu
225 230 235 240
Ala Val Asp Lys Ala Ser Trp Gln Arg Gly Asp Thr Phe Gln Cys Ala
245 250 255
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Ile Phe
260 265 270
Lys Thr Pro Gly Lys
275
<210> 105
<211> 834
<212> ДНК
<213> Bubalus arnee
<400> 105
accttcccat ccgtcctgca gccgtcaggg ctctactccc tcagcagcat ggtgaccgtg 60
ccggccagca gcctgtccag caagagctac acctgcaatg tcaaccaccc ggccaccacc 120
accaaggtgg acaagcgtgt tggaacaaag accaaaccac catgtcccat atgcccagcc 180
tgtgaagggc ccgggccctc ggccttcatc ttccctccaa aacccaagga caccctcatg 240
atctcccgga cccccaaggt cacgtgcgtg gtggtagatg tgagccagga gaacccggag 300
gtccagttct cctggtacgt ggacggcgta gaggtgcaca cggcccagac gaggccaaag 360
gaggagcagt tcaacagcac ctaccgcgtg gtcagcgtcc tgcccatcca gcaccaggac 420
tggctgaacg ggaaggagtt caagtgcaag gtcaacaaca aagacctccc agcccccatc 480
acaaggatca tctccaaggc caaagggcag acccgggagc cgcaggtgta caccctgccc 540
ccacccaccg aggagctgtc caggagcaaa gtcacgctaa cctgcctggt cactggcttc 600
tacccacctg acatcgatgt cgagtggcaa agaaacggac agccggagcc agagggcaat 660
taccgcacca ccccgcccca gcaggacgtg gacgggacct acttcctgta cagcaagctc 720
gcggtggaca aggccagctg gcagcgtgga gacacattcc agtgtgcggt gatgcacgag 780
gctctgcaca accactacac ccagaagtcc atcttcaaga ctccgggtaa atga 834
<210> 106
<211> 318
<212> Белок
<213> Bubalus arnee
<400> 106
Ala Pro Lys Thr Ala Pro Ser Val Tyr Pro Leu Ala Pro Cys Gly Arg
1 5 10 15
Asp Val Ser Gly Pro Asn Val Ala Leu Gly Cys Leu Ala Ser Ser Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Thr Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ser Val Leu Gln Pro Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Met Val Thr Val Pro Ala Ser Ser Leu Ser Ser Lys Ser
65 70 75 80
Tyr Thr Cys Asn Val Asn His Pro Ala Thr Thr Thr Lys Val Asp Lys
85 90 95
Arg Val Gly Ile His Gln Pro Gln Thr Cys Pro Ile Cys Pro Ala Cys
100 105 110
Glu Gly Pro Gly Pro Ser Ala Phe Ile Phe Pro Pro Lys Pro Lys Asp
115 120 125
Thr Leu Met Ile Ser Arg Thr Pro Lys Val Thr Cys Val Val Val Asp
130 135 140
Val Ser Gln Glu Asn Pro Glu Val Gln Phe Ser Trp Tyr Val Asp Gly
145 150 155 160
Val Glu Val His Thr Ala Gln Thr Arg Pro Lys Glu Glu Gln Phe Asn
165 170 175
Ser Thr Tyr Arg Val Val Ser Val Leu Leu Ile Gln His Gln Asp Trp
180 185 190
Leu Asn Gly Lys Glu Phe Lys Cys Lys Val Asn Asn Lys Asp Leu Pro
195 200 205
Ala Pro Ile Thr Arg Ile Ile Ser Lys Ala Lys Gly Gln Thr Arg Glu
210 215 220
Pro Gln Val Tyr Thr Leu Pro Pro Pro Thr Glu Glu Leu Ser Arg Ser
225 230 235 240
Lys Val Thr Leu Thr Cys Leu Val Thr Gly Phe Tyr Pro Pro Asp Ile
245 250 255
Asp Val Glu Trp Gln Arg Asn Gly Gln Pro Glu Pro Glu Gly Asn Tyr
260 265 270
Arg Thr Thr Pro Pro Gln Gln Asp Val Asp Gly Thr Tyr Phe Leu Tyr
275 280 285
Ser Lys Leu Ala Val Asp Lys Ala Ser Trp Gln Arg Gly Asp Thr Phe
290 295 300
Gln Cys Ala Val Met His Glu Ala Leu His Asn His Tyr Thr
305 310 315
<210> 107
<211> 955
<212> ДНК
<213> Bubalus arnee
<400> 107
gcccccaaga cggccccatc ggtctaccct ctggccccct gcggcaggga cgtgtctggc 60
cctaacgtgg ccttgggctg cctggcctca agctacttcc ccgagccagt gaccgtgacc 120
tggaactcgg gcgccctgac cagtggcgtg cacaccttcc catccgtcct gcagccgtca 180
gggctctact ccctcagcag catggtgacc gtgccggcca gcagcctgtc cagcaagagc 240
tacacctgca atgtcaacca cccggccacc accaccaagg tggacaagcg tgttggaata 300
caccagccgc aaacatgtcc catatgccca gcctgtgaag ggcccgggcc ctcggccttc 360
atcttccctc caaaacccaa ggacaccctc atgatctccc ggacccccaa ggtcacgtgc 420
gtggtggttg atgtgagcca ggagaacccg gaggtccagt tctcctggta cgtggacggc 480
gtagaggtgc acacggccca gacgaggcca aaggaggagc agttcaacag cacctaccgc 540
gtggtcagcg tcctgctcat ccagcaccag gactggctga acgggaagga gttcaagtgc 600
aaggtcaaca acaaagacct cccagccccc atcacaagga tcatctccaa ggccaaaggg 660
cagacccggg agccgcaggt gtacaccctg cccccaccca ccgaggagct gtccaggagc 720
aaagtcacgc taacctgcct ggtcactggc ttctacccac ctgacatcga tgtcgagtgg 780
caaagaaacg gacagccgga gccagagggc aattaccgca ccaccccgcc ccagcaggac 840
gtggacggga cctacttcct gtacagcaag ctcgcggtgg acaaggccag ctggcagcgt 900
ggagacacat tccagtgtgc ggtgatgcac gaggctctgc acaaccacta caccc 955
<210> 108
<211> 323
<212> Белок
<213> Bubalus arnee
<400> 108
Ala Pro Lys Thr Ala Pro Ser Val Tyr Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Asp Thr Ser Gly Pro Asn Val Ala Leu Gly Cys Leu Val Ser Ser Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Thr Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ser Val Leu Gln Pro Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Met Val Thr Val Pro Ala His Ser Leu Ser Ser Lys Arg
65 70 75 80
Tyr Thr Cys Asn Val Asn His Pro Ala Thr Lys Thr Lys Val Asp Leu
85 90 95
Cys Val Gly Arg Pro Cys Pro Ile Cys Pro Gly Cys Glu Val Ala Gly
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Lys Pro Lys Asp Ile Leu Met Ile
115 120 125
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser Lys Glu
130 135 140
His Ala Glu Val Gln Phe Ser Trp Tyr Val Asp Gly Glu Glu Val His
145 150 155 160
Thr Ala Glu Thr Arg Pro Lys Glu Glu Gln Phe Asn Ser Thr Tyr Arg
165 170 175
Val Val Ser Val Leu Pro Ile Gln His Glu Asp Trp Leu Lys Gly Lys
180 185 190
Glu Phe Glu Cys Lys Val Asn Asn Glu Asp Leu Pro Gly Pro Ile Thr
195 200 205
Arg Thr Ile Ser Lys Ala Lys Gly Val Val Arg Ser Pro Glu Val Tyr
210 215 220
Thr Leu Pro Pro Pro Ala Glu Glu Leu Ser Lys Ser Ile Val Thr Leu
225 230 235 240
Thr Cys Leu Val Lys Ser Ile Phe Pro Phe Ile His Val Glu Trp Lys
245 250 255
Ile Asn Gly Lys Pro Glu Pro Glu Asn Ala Tyr Arg Thr Thr Pro Pro
260 265 270
Gln Glu Asp Glu Asp Arg Thr Tyr Phe Leu Tyr Ser Lys Leu Ala Val
275 280 285
Asp Lys Ala Arg Trp Asp His Gly Glu Thr Phe Glu Cys Ala Val Met
290 295 300
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Ile Ser Lys Thr
305 310 315 320
Gln Gly Lys
<210> 109
<211> 975
<212> ДНК
<213> Bubalus arnee
<220>
<221> misc_feature
<222> (748)..(748)
<223> n is a, c, g, or t
<400> 109
gcccccaaga cggccccatc ggtctaccct ctggccccct gcagcaggga cacgtctggc 60
cctaacgtgg ccttgggctg cctggtctca agctacttcc ccgagccagt gaccgtgacc 120
tggaactcgg gcgccctgac cagtggcgtg cacaccttcc catccgtcct gcagccgtca 180
gggctctact ccctcagcag catggtgacc gtgccggccc acagcttgtc cagcaagcgc 240
tatacgtgca atgtcaacca cccagccacc aaaaccaagg tggacctgtg tgttggacga 300
ccatgtccca tatgcccagg ctgtgaagtg gccgggccct cggtcttcat cttccctcca 360
aaacccaagg acatcctcat gatctcccgg acccccgagg tcacgtgcgt ggtggtggac 420
gtcagcaagg agcacgccga ggtccagttc tcctggtacg tggacggcga agaggtgcac 480
acggccgaga cgaggccaaa ggaggagcag ttcaacagca cctaccgcgt ggtcagcgtc 540
ctgcccatcc agcacgagga ctggctgaag gggaaggagt tcgagtgcaa ggtcaacaac 600
gaagacctcc caggccccat cacgaggacc atctccaagg ccaaaggggt ggtacggagc 660
ccggaggtgt acaccctgcc cccacccgcc gaggagctgt ccaagagcat agtcacgcta 720
acctgcctgg tcaaaagcat cttcccgnct ttcatccatg ttgagtggaa aatcaacgga 780
aaaccagagc cagagaacgc atatcgcacc accccgcctc aggaggacga ggacaggacc 840
tacttcctgt acagcaagct cgcggtggac aaggcaagat gggaccatgg agaaacattt 900
gagtgtgcgg tgatgcacga ggctctgcac aaccactaca cccagaagtc catctccaag 960
actcagggta aatga 975
<210> 110
<211> 317
<212> Белок
<213> Bubalus arnee
<400> 110
Ala Tyr Asn Thr Ala Pro Ser Val Tyr Pro Leu Ala Pro Cys Gly Arg
1 5 10 15
Asp Val Ser Asp His Asn Val Ala Leu Gly Cys Leu Val Ser Ser Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Thr Trp Asn Trp Gly Ala Gln Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ser Val Leu Gln Pro Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Thr Val Thr Val Pro Ala His Ser Leu Ser Ser Lys Cys
65 70 75 80
Phe Thr Cys Asn Val Asn His Pro Ala Thr Thr Thr Lys Val Asp Leu
85 90 95
Cys Val Gly Lys Lys Thr Lys Pro Arg Cys Pro Ile Cys Pro Gly Cys
100 105 110
Glu Val Ala Gly Pro Ser Val Phe Ile Phe Pro Pro Lys Pro Lys Asp
115 120 125
Ile Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
130 135 140
Val Ser Lys Glu His Ala Glu Val Gln Phe Ser Trp Tyr Val Asp Gly
145 150 155 160
Glu Glu Val His Thr Ala Glu Thr Arg Pro Lys Glu Glu Gln Phe Asn
165 170 175
Ser Thr Tyr Arg Val Val Ser Val Leu Pro Ile Gln His Glu Asp Trp
180 185 190
Leu Lys Gly Lys Glu Phe Glu Cys Lys Val Asn Asn Glu Asp Leu Pro
195 200 205
Gly Pro Ile Thr Arg Thr Ile Ser Lys Ala Lys Gly Val Val Arg Ser
210 215 220
Pro Glu Val Tyr Thr Leu Pro Pro Pro Ala Glu Glu Leu Ser Lys Ser
225 230 235 240
Ile Val Thr Leu Thr Cys Leu Val Lys Ser Phe Phe Pro Pro Phe Ile
245 250 255
His Val Glu Trp Lys Ile Asn Gly Lys Pro Glu Pro Glu Asn Ala Tyr
260 265 270
Arg Thr Thr Pro Pro Gln Glu Asp Glu Asp Gly Thr Tyr Phe Leu Tyr
275 280 285
Ser Lys Phe Ser Val Glu Lys Phe Arg Trp His Ser Gly Gly Ile His
290 295 300
Cys Ala Val Met His Glu Ala Leu His Asn His Tyr Thr
305 310 315
<210> 111
<211> 952
<212> ДНК
<213> Bubalus arnee
<400> 111
gcctacaaca cagctccatc ggtctaccct ctggccccct gtggcaggga cgtgtctgat 60
cataacgtgg ccttgggctg cctggtctca agctacttcc ccgagccagt gaccgtgacc 120
tggaactggg gcgcccagac cagtggcgtg cacaccttcc catccgtcct gcagccgtca 180
gggctctact ccctcagcag cacggtgacc gtgccggccc acagcttgtc cagcaagtgc 240
ttcacgtgca atgtcaacca cccggccacc accaccaagg tggacctgtg tgttggaaaa 300
aagaccaagc ctcgatgtcc catatgccca ggctgtgaag tggccgggcc ctcggtcttc 360
atcttccctc caaaacccaa ggacatcctc atgatctccc ggacccccga ggtcacgtgc 420
gtggtggtgg acgtcagcaa ggagcacgcc gaggtccagt tctcctggta cgtggacggc 480
gaagaggtgc acacggccga gacgagacca aaggaggagc agttcaacag cacttaccgc 540
gtggtcagcg tcctgcccat ccagcacgag gactggctga aggggaagga gttcgagtgc 600
aaggtcaaca acgaagacct cccaggcccc atcacgagga ccatctccaa ggccaaaggg 660
gtggtacgga gcccggaggt gtacaccctg cccccacccg ccgaggagct gtccaagagc 720
atagtcacgc taacctgcct ggtcaaaagc ttcttcccgc ctttcatcca tgttgagtgg 780
aaaatcaacg gaaaaccaga gccagagaac gcataccgca ccaccccgcc ccaggaggac 840
gaggacggga cctacttcct gtacagcaag ttctcggtgg aaaagttcag gtggcacagt 900
ggaggcatcc actgtgcggt gatgcacgag gctctgcaca accactacac cc 952
<210> 112
<211> 314
<212> Белок
<213> Bubalus arnee
<400> 112
Ala Pro Lys Thr Ala Pro Ser Val Tyr Pro Leu Ala Pro Cys Gly Arg
1 5 10 15
Asp Thr Ser Gly Pro Asn Val Ala Leu Gly Cys Leu Ala Ser Ser Tyr
20 25 30
Phe Pro Glu Pro Val Thr Leu Thr Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ser Val Leu Gln Pro Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Met Val Thr Val Pro Ala Ser Ser Leu Ser Ser Lys Ser
65 70 75 80
Tyr Thr Cys Asn Val Asn His Pro Ala Thr Thr Thr Lys Val Asp Leu
85 90 95
Cys Val Gly Arg Pro Cys Pro Ile Cys Pro Ala Cys Glu Gly Pro Gly
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
115 120 125
Ser Arg Thr Pro Gln Val Thr Cys Val Val Val Asp Val Ser Gln Glu
130 135 140
Asn Pro Glu Val Gln Phe Ser Trp Tyr Val Asp Gly Val Glu Val His
145 150 155 160
Thr Ala Gln Thr Arg Pro Lys Glu Ala Gln Phe Asn Ser Thr Tyr Arg
165 170 175
Val Val Ser Val Leu Pro Ile Gln His Glu Asp Trp Leu Lys Gly Lys
180 185 190
Glu Phe Glu Cys Lys Val Asn Asn Lys Asp Leu Pro Ala Pro Ile Thr
195 200 205
Arg Ile Ile Ser Lys Ala Lys Gly Pro Ser Arg Glu Pro Gln Val Tyr
210 215 220
Thr Leu Ser Pro Ser Ala Glu Glu Leu Ser Arg Ser Lys Val Ser Ile
225 230 235 240
Thr Cys Leu Val Thr Gly Phe Tyr Pro Pro Asp Ile Asp Val Glu Trp
245 250 255
Lys Ser Asn Gly Gln Pro Glu Pro Glu Gly Asn Tyr Arg Thr Thr Pro
260 265 270
Pro Gln Gln Asp Val Asp Gly Thr Tyr Phe Leu Tyr Ser Lys Leu Ala
275 280 285
Val Asp Lys Ala Ser Trp Gln Arg Gly Asp Pro Phe Gln Cys Ala Val
290 295 300
Met His Glu Ala Leu His Asn His Tyr Thr
305 310
<210> 113
<211> 943
<212> ДНК
<213> Bubalus arnee
<400> 113
gcccccaaga cggccccatc ggtctaccct ctggccccct gcggcaggga cacgtctggc 60
cctaacgtgg ccttgggctg cctggcctca agctacttcc ccgagccagt gaccctgacc 120
tggaactcgg gcgccctgac cagtggcgtg cataccttcc catccgtcct gcagccgtca 180
gggctctact ccctcagcag catggtgacc gtgccggcca gcagcctgtc cagcaagagc 240
tacacctgca atgtcaacca cccggccacc accaccaagg tggacctgtg tgttggacga 300
ccatgtccca tatgcccagc ctgtgaaggg cccgggccct cggtcttcat cttccctcca 360
aaacccaagg acaccctcat gatctcccgg acaccccagg tcacgtgcgt ggtggtagat 420
gtgagccagg aaaacccgga ggtccagttc tcctggtatg tggacggtgt agaggtgcac 480
acggcccaga cgaggccaaa ggaggcgcag ttcaacagca cctaccgtgt ggtcagcgtc 540
ctgcccatcc agcacgagga ctggctgaag gggaaggagt tcgagtgcaa ggtcaacaac 600
aaagacctcc cagcccccat cacaaggatc atctccaagg ccaaagggcc gagccgggag 660
ccgcaggtgt acaccctgtc cccatccgcc gaggagctgt ccaggagcaa agtcagcata 720
acctgcctgg tcactggctt ctacccacct gacatcgatg tcgagtggaa gagcaacgga 780
cagccggagc cagagggcaa ttaccgcacc accccgcccc agcaggacgt ggacgggacc 840
tacttcctgt acagcaagct cgcggtggac aaggccagct ggcagcgtgg agacccattc 900
cagtgtgcgg tgatgcacga ggctctgcac aaccactaca ccc 943
<210> 114
<211> 320
<212> Белок
<213> Bubalus arnee
<400> 114
Ala Pro Lys Thr Ala Pro Ser Val Tyr Pro Leu Ala Pro Cys Gly Arg
1 5 10 15
Asp Thr Ser Gly Pro Asn Val Ala Leu Gly Cys Leu Ala Ser Ser Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Thr Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ser Val Leu Gln Pro Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Thr Val Thr Val Pro Ala Arg Ser Ser Ser Arg Lys Cys
65 70 75 80
Phe Thr Cys Asn Val Asn His Pro Ala Thr Thr Thr Lys Val Asp Leu
85 90 95
Cys Val Gly Arg Pro Cys Pro Ile Cys Pro Ala Cys Glu Gly Asn Gly
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
115 120 125
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu
130 135 140
Asn Pro Glu Val Gln Phe Ser Trp Tyr Val Asp Gly Glu Glu Val His
145 150 155 160
Thr Ala Glu Thr Arg Pro Lys Glu Glu Gln Phe Asn Ser Thr Tyr Arg
165 170 175
Val Val Ser Val Leu Pro Ile Gln His Gln Asp Trp Leu Lys Gly Lys
180 185 190
Glu Phe Glu Cys Lys Val Asn Asn Lys Asp Leu Pro Ala Pro Ile Thr
195 200 205
Arg Ile Ile Ser Lys Ala Lys Gly Pro Ser Arg Glu Pro Gln Val Tyr
210 215 220
Thr Leu Ser Pro Ser Ala Glu Glu Leu Ser Arg Ser Lys Val Ser Ile
225 230 235 240
Thr Cys Leu Val Thr Gly Phe Tyr Pro Pro Asp Ile Asp Val Glu Trp
245 250 255
Lys Ser Asn Gly Gln Pro Glu Pro Glu Gly Asn Tyr Arg Ser Thr Pro
260 265 270
Pro Gln Glu Asp Glu Asp Gly Thr Tyr Phe Leu Tyr Ser Lys Leu Ala
275 280 285
Val Asp Lys Ala Arg Leu Gln Ser Gly Gly Ile His Cys Ala Val Met
290 295 300
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Ile Ser Lys Thr
305 310 315 320
<210> 115
<211> 960
<212> ДНК
<213> Bubalus arnee
<400> 115
gcccccaaga cggccccatc ggtctaccct ctggccccct gcggcaggga cacgtctggc 60
cctaacgtgg ccttgggctg cctggcctca agctacttcc ccgagccagt gaccgtgacc 120
tggaactcgg gcgccctgac cagtggcgtg cacaccttcc catccgtcct gcagccgtca 180
gggctctact ccctcagcag cacggtgacc gtgccggcca ggagctcgtc cagaaagtgc 240
ttcacgtgca atgtcaacca cccggccacc accaccaagg tggacctgtg tgttggacga 300
ccatgtccca tatgcccagc ctgtgaaggg aacgggccct cggtcttcat cttccctcca 360
aaacccaagg acaccctcat gatctcccgg acccccgagg tcacgtgcgt ggtggtagat 420
gtgagccagg aaaacccgga ggtccagttc tcctggtacg tggacggcga agaggtgcac 480
acggccgaga cgaggccaaa ggaggagcag ttcaacagca cctaccgtgt ggtcagcgtc 540
ctgcccatcc agcaccagga ctggctgaag ggaaaggagt tcgagtgcaa ggtcaacaac 600
aaagacctcc cagcccccat cacaaggatc atctccaagg ccaaagggcc gagccgggag 660
ccgcaggtgt acaccctgtc cccatccgcc gaggagctgt ccaggagcaa agtcagcata 720
acctgcctgg tcactggctt ctacccacct gacatcgatg tcgagtggaa gagcaacgga 780
cagccggagc cagagggcaa ttaccgctcc accccgcccc aggaggacga ggacgggacc 840
tacttcctgt acagcaaact cgcggtggac aaggcgaggt tgcagagtgg aggcatccac 900
tgtgcggtga tgcacgaggc tctgcacaac cactacaccc agaagtccat ctccaagact 960
<210> 116
<211> 105
<212> Белок
<213> Bubalus arnee
<400> 116
Gln Pro Lys Ser Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Thr Glu
1 5 10 15
Glu Leu Ser Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe
20 25 30
Tyr Pro Gly Ser Met Thr Val Ala Arg Lys Ala Asp Gly Ser Thr Ile
35 40 45
Thr Arg Asn Val Glu Thr Thr Arg Ala Ser Lys Gln Ser Asn Ser Lys
50 55 60
Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Gly Ser Glu Trp Lys Ser
65 70 75 80
Lys Gly Ser Tyr Ser Cys Glu Val Thr His Glu Gly Ser Thr Val Thr
85 90 95
Lys Thr Val Lys Pro Ser Glu Cys Ser
100 105
<210> 117
<211> 318
<212> ДНК
<213> Bubalus arnee
<400> 117
cagcccaagt ccgcaccctc agtcaccctg ttcccaccct ccacggagga gctcagcgcc 60
aacaaggcca ccctggtgtg tctcatcagc gacttctacc cgggtagcat gaccgtggcc 120
aggaaggcag acggcagcac catcacccgg aacgtggaga ccacccgggc ctccaaacag 180
agcaacagca agtacgcggc cagcagctac ctgagcctga cgggcagcga gtggaaatcg 240
aaaggcagtt acagctgcga ggtcacgcac gaggggagca ccgtgacaaa gacagtgaag 300
ccctcagagt gttcttag 318
<210> 118
<211> 266
<212> Белок
<213> Bubalus arnee
<400> 118
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
1 5 10 15
Ser Leu Ser Ser Thr Val Thr Ala Pro Ala Ser Ala Thr Lys Ser Gln
20 25 30
Thr Phe Thr Cys Asn Val Ala His Pro Ala Ser Ser Thr Lys Val Asp
35 40 45
Lys Ala Val Val Pro Pro Cys Arg Pro Lys Pro Cys Asp Cys Cys Pro
50 55 60
Pro Pro Glu Leu Pro Gly Gly Pro Ser Val Phe Ile Phe Pro Pro Lys
65 70 75 80
Pro Lys Asp Thr Leu Thr Ile Ser Gly Thr Pro Glu Val Thr Cys Val
85 90 95
Val Val Asp Val Gly His Asp Asp Pro Glu Val Lys Phe Ser Trp Phe
100 105 110
Val Asp Asp Val Glu Val Asn Thr Ala Arg Thr Lys Pro Arg Glu Glu
115 120 125
Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Ala Leu Pro Ile Gln His
130 135 140
Asn Asp Trp Thr Gly Gly Lys Glu Phe Lys Cys Lys Val Tyr Asn Glu
145 150 155 160
Gly Leu Pro Ala Pro Ile Val Arg Thr Ile Ser Arg Thr Lys Gly Gln
165 170 175
Ala Arg Glu Pro Gln Val Tyr Val Leu Ala Pro Pro Gln Asp Glu Leu
180 185 190
Ser Lys Ser Thr Val Ser Ile Thr Cys Met Val Thr Gly Phe Tyr Pro
195 200 205
Asp Tyr Ile Ala Val Glu Trp Gln Lys Asp Gly Gln Pro Glu Ser Glu
210 215 220
Asp Lys Tyr Gly Thr Thr Pro Pro Gln Leu Asp Ser Asp Gly Ser Tyr
225 230 235 240
Phe Leu Tyr Ser Arg Leu Arg Val Asn Lys Asn Ser Trp Gln Glu Gly
245 250 255
Gly Ala Tyr Thr Cys Val Val Met His Glu
260 265
<210> 119
<211> 801
<212> ДНК
<213> Bubalus arnee
<400> 119
gagcggcgtg cacaccttcc cggccgtcct tcagtcctcc gggctctact ctctcagcag 60
cacggtgacc gcgcccgcca gcgccacaaa aagccagacc ttcacctgca acgtagccca 120
cccggccagc agcaccaagg tggacaaggc tgttgttccc ccatgcagac cgaaaccctg 180
tgattgctgc ccaccccctg agctccccgg aggaccctct gtcttcatct tcccaccaaa 240
acccaaggac accctcacaa tctctggaac tcctgaggtc acgtgtgtgg tggtggacgt 300
gggccacgat gaccccgagg tgaagttctc ctggttcgtg gacgatgtgg aggtaaacac 360
agccaggacg aagccaagag aggagcagtt caacagcacc taccgcgtgg tcagcgccct 420
gcccatccag cacaacgact ggactggagg aaaggagttc aagtgcaagg tctacaatga 480
aggcctccca gcccccatcg tgaggaccat ctccaggacc aaagggcagg cccgggagcc 540
gcaggtgtac gtcctggccc caccccagga cgagctcagc aaaagcacgg tcagcatcac 600
ttgcatggtc actggcttct acccagacta catcgccgta gagtggcaga aagatgggca 660
gcctgagtca gaggacaaat atggcacgac cccgccccag ctggacagcg atggctccta 720
cttcctgtac agcaggctca gggtgaacaa gaacagctgg caagaaggag gcgcctacac 780
gtgtgtagtg atgcatgagg c 801
<210> 120
<211> 309
<212> Белок
<213> Bubalus arnee
<400> 120
Ala Ser Ile Thr Ala Pro Lys Val Tyr Pro Leu Thr Ser Cys Arg Gly
1 5 10 15
Glu Thr Ser Ser Ser Thr Val Thr Leu Gly Cys Leu Val Ser Ser Tyr
20 25 30
Met Pro Glu Pro Val Thr Val Thr Trp Asn Ser Gly Ala Leu Lys Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Thr Val Thr Ala Pro Ala Ser Ala Thr Lys Ser Gln Thr
65 70 75 80
Phe Thr Cys Asn Val Ala His Pro Ala Ser Ser Thr Lys Val Asp Thr
85 90 95
Ala Val Gly Phe Ser Ser Asp Cys Cys Lys Phe Pro Lys Pro Cys Val
100 105 110
Arg Gly Pro Ser Val Phe Ile Phe Pro Pro Lys Pro Lys Asp Thr Leu
115 120 125
Met Ile Thr Gly Asn Pro Glu Val Thr Cys Val Val Val Asp Val Gly
130 135 140
Arg Asp Asn Pro Glu Val Gln Phe Ser Trp Phe Val Gly Asp Val Glu
145 150 155 160
Val His Thr Gly Arg Ser Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr
165 170 175
Tyr Arg Val Val Ser Thr Leu Pro Ile Gln His Asn Asp Trp Thr Gly
180 185 190
Gly Lys Glu Phe Lys Cys Lys Val Asn Asn Lys Gly Leu Pro Ala Pro
195 200 205
Ile Val Arg Thr Ile Ser Arg Thr Lys Gly Gln Ala Arg Glu Pro Gln
210 215 220
Val Tyr Val Leu Ala Pro Pro Gln Glu Glu Leu Ser Lys Ser Thr Val
225 230 235 240
Ser Val Thr Cys Met Val Thr Gly Phe Tyr Pro Asp Tyr Ile Ala Val
245 250 255
Glu Trp His Arg Asp Arg Gln Ala Glu Ser Glu Asp Lys Tyr Arg Thr
260 265 270
Thr Pro Pro Gln Leu Asp Ser Asp Gly Ser Tyr Phe Leu Tyr Ser Arg
275 280 285
Leu Lys Val Asn Lys Asn Ser Trp Gln Glu Gly Gly Ala Tyr Thr Cys
290 295 300
Val Val Met His Glu
305
<210> 121
<211> 929
<212> ДНК
<213> Bubalus arnee
<400> 121
gcctccatca cagccccgaa agtctaccct ctgacttctt gccgcgggga aacgtccagc 60
tccaccgtga ccctgggctg cctggtctcc agctacatgc ccgagccggt gaccgtgacc 120
tggaactcgg gtgccctgaa gagcggcgtg cacaccttcc cggccgtcct tcagtcctct 180
gggctctact ctctcagcag cacggtgacc gcgcccgcca gcgccacaaa aagccagacc 240
ttcacctgca acgtagccca cccggccagc agcaccaagg tggacacggc tgttgggttc 300
tccagtgact gctgcaagtt tcctaagcct tgtgtgaggg gaccatctgt cttcatcttc 360
ccgccgaaac ccaaagacac cctgatgatc acaggaaatc ccgaggtcac atgtgtggtg 420
gtggacgtgg gccgggataa ccccgaggtg cagttctcct ggttcgtggg tgatgtggag 480
gtgcacacgg gcaggtcgaa gccgagagag gagcagttca acagcaccta ccgcgtggtc 540
agcaccctgc ccatccagca caatgactgg actggaggaa aggagttcaa gtgcaaggtc 600
aacaacaaag gcctcccagc ccccatcgtg aggaccatct ccaggaccaa agggcaggcc 660
cgggagccgc aggtgtacgt cctggcccca ccccaggaag agctcagcaa aagcacggtc 720
agcgtcactt gcatggtcac tggcttctac ccagactaca tcgccgtaga gtggcataga 780
gaccggcagg ctgagtcgga ggacaagtac cgcacgaccc cgccccagct ggacagcgat 840
ggctcctact tcctgtacag caggctcaag gtgaacaaga acagctggca agaaggaggc 900
gcctacacgt gtgtagtgat gcatgaggc 929
<210> 122
<211> 352
<212> Белок
<213> Bubalus arnee
<400> 122
Ala Ser Thr Thr Ala Pro Lys Val Tyr Pro Leu Ala Ser Ser Cys Gly
1 5 10 15
Asp Thr Ser Ser Ser Thr Val Thr Leu Gly Cys Leu Val Ser Ser Tyr
20 25 30
Met Pro Glu Pro Val Thr Val Thr Trp Asn Ser Gly Ala Leu Lys Asn
35 40 45
Gly Val His Thr Phe Pro Ala Val Arg Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Met Val Thr Met Pro Thr Ser Thr Ala Gly Thr Gln Thr
65 70 75 80
Phe Thr Cys Asn Val Ala His Pro Ala Ser Ser Thr Lys Val Asp Thr
85 90 95
Ala Val Thr Ala Arg His Pro Val Pro Lys Thr Pro Glu Thr Pro Ile
100 105 110
His Pro Val Lys Pro Pro Thr Gln Glu Pro Arg Asp Glu Lys Thr Pro
115 120 125
Cys Gln Cys Pro Lys Cys Pro Glu Pro Leu Gly Gly Leu Ser Val Phe
130 135 140
Ile Phe Pro Pro Lys Pro Lys Asp Thr Leu Thr Ile Ser Gly Thr Pro
145 150 155 160
Glu Val Thr Cys Val Val Val Asp Val Gly Gln Asp Asp Pro Glu Val
165 170 175
Gln Phe Ser Trp Phe Val Asp Asp Val Glu Val His Thr Ala Arg Met
180 185 190
Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Ala
195 200 205
Leu Pro Ile Gln His Gln Asp Trp Leu Arg Glu Lys Glu Phe Lys Cys
210 215 220
Lys Val Asn Asn Lys Gly Leu Pro Ala Pro Ile Val Arg Thr Ile Ser
225 230 235 240
Arg Thr Lys Gly Gln Ala Arg Glu Pro Gln Val Tyr Val Leu Ala Pro
245 250 255
Pro Arg Glu Glu Leu Ser Lys Ser Thr Leu Ser Leu Thr Cys Leu Ile
260 265 270
Thr Gly Phe Tyr Pro Glu Glu Val Asp Val Glu Trp Gln Arg Asn Gly
275 280 285
Gln Pro Glu Ser Glu Asp Lys Tyr His Thr Thr Pro Pro Gln Leu Asp
290 295 300
Ala Asp Gly Ser Tyr Phe Leu Tyr Ser Arg Leu Arg Val Asn Arg Ser
305 310 315 320
Ser Trp Gln Glu Gly Asp His Tyr Thr Cys Ala Val Met His Glu Ala
325 330 335
Leu Arg Asn His Tyr Lys Glu Lys Pro Ile Ser Arg Ser Pro Gly Lys
340 345 350
<210> 123
<211> 1059
<212> ДНК
<213> Bubalus bubalis
<400> 123
gcctccacca cagccccgaa agtctaccct ctggcatcca gctgcgggga cacgtccagc 60
tccaccgtga ccctgggctg cctggtctcc agctacatgc ccgagccggt gaccgtgacc 120
tggaactcgg gtgccctgaa gaacggcgtg cacaccttcc cggccgtccg gcagtcctcc 180
gggctctact ctctcagcag catggtgacc atgcccacca gcaccgcagg aacccagacc 240
ttcacctgca acgtagccca cccggccagc agcaccaagg tggacacggc tgtcactgca 300
aggcatccgg tcccgaagac accagagaca cctatccatc ctgtaaaacc cccaacccag 360
gagcccagag atgaaaagac accctgccag tgtcccaaat gcccagaacc tctgggagga 420
ctgtctgtct tcatcttccc accgaaaccc aaggacaccc tcacaatctc tggaacgccc 480
gaggtcacgt gtgtggtggt ggacgtgggc caggatgacc ccgaagtgca gttctcctgg 540
ttcgtggatg acgtggaggt gcacacagcc aggatgaagc caagagagga gcagttcaac 600
agcacctacc gcgtggtcag cgccctgccc atccagcacc aggactggct gcgggaaaag 660
gagttcaagt gcaaggtcaa caacaaaggc ctcccggccc ccatcgtgag gaccatctcc 720
aggaccaaag ggcaggcccg ggagccacag gtgtatgtcc tggccccacc ccgggaagag 780
ctcagcaaaa gcacgctcag cctcacctgc ctaatcaccg gcttctaccc agaagaggta 840
gacgtggagt ggcagagaaa tgggcagcct gagtcagagg acaagtacca cacgacccca 900
ccccagctgg acgctgacgg ctcctacttc ctgtacagca ggctcagggt gaacaggagc 960
agctggcagg aaggagacca ctacacgtgt gcagtgatgc atgaagcttt acggaatcac 1020
tacaaagaga agcccatctc gaggtctccg ggtaaatga 1059
<210> 124
<211> 35
<212> ДНК
<213> Искусственная последовательность
<220>
<223> primer
<400> 124
ctagctagca ccatgaggat atatagtgtc ttaac 35
<210> 125
<211> 31
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 125
caatctcgag ttacagacag aagatgactg c 31
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| АНТИТЕЛА К PD-L1, СВЯЗЫВАЮЩИЕ PD-L1 СОБАКИ | 2015 |
|
RU2722562C2 |
| АНТИТЕЛА К PD-L1, СВЯЗЫВАЮЩИЕ PD-L1 СОБАКИ | 2015 |
|
RU2832918C2 |
| PD-L1 специфические антитела | 2017 |
|
RU2756236C2 |
| СВЯЗЫВАЮЩИЕСЯ С CD38 И PD-L1 МОЛЕКУЛЫ | 2017 |
|
RU2764201C2 |
| АНТИТЕЛА ПРОТИВ PD-L1 И ИХ ВАРИАНТЫ | 2017 |
|
RU2770590C2 |
| СВЯЗЫВАЮЩИЕ МОЛЕКУЛЫ, СВЯЗЫВАЮЩИЕ PD-L1 и LAG-3 | 2017 |
|
RU2784388C2 |
| Анти-PD-L1 и IL-2 цитокины | 2017 |
|
RU2769282C2 |
| НОВЫЙ СЛИТЫЙ БЕЛОК, СПЕЦИФИЧНЫЙ ДЛЯ CD137 И PD-L1 | 2019 |
|
RU2818349C2 |
| МОЛЕКУЛЫ АНТИТЕЛА, КОТОРЫЕ СВЯЗЫВАЮТ PD-L1 и CD137 | 2019 |
|
RU2826084C2 |
| СПЕЦИФИЧЕСКИЕ АНТИТЕЛА К PD-L1 И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2017 |
|
RU2749109C2 |



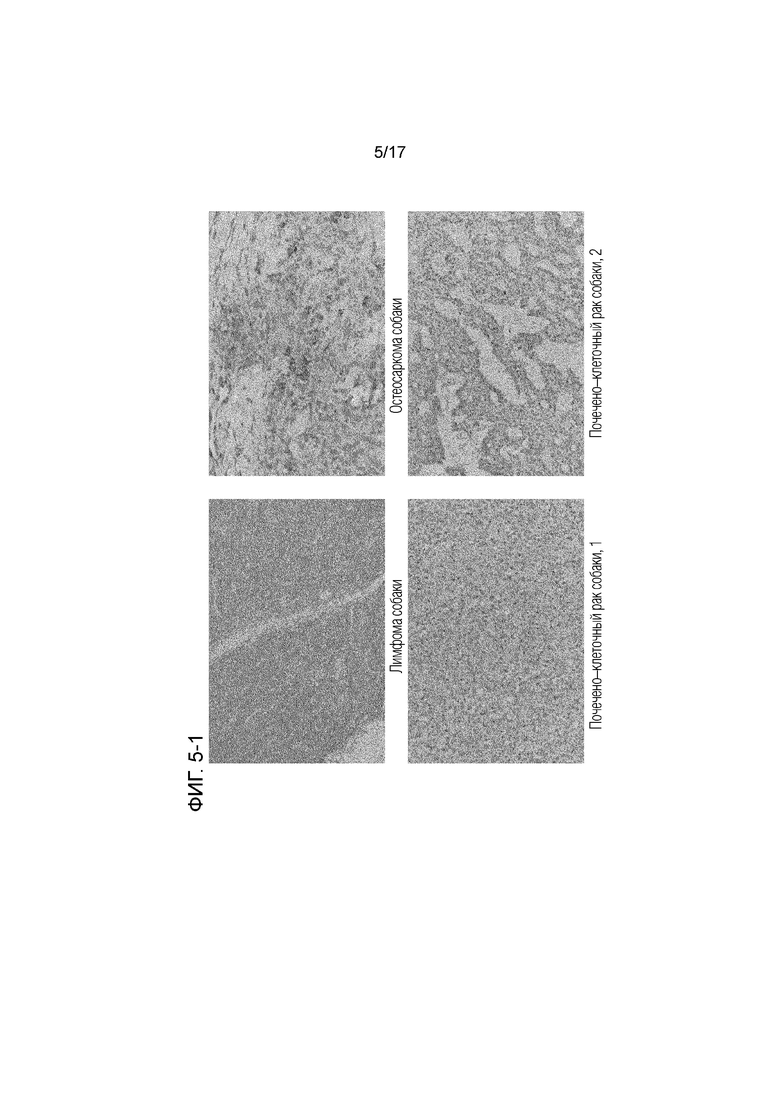
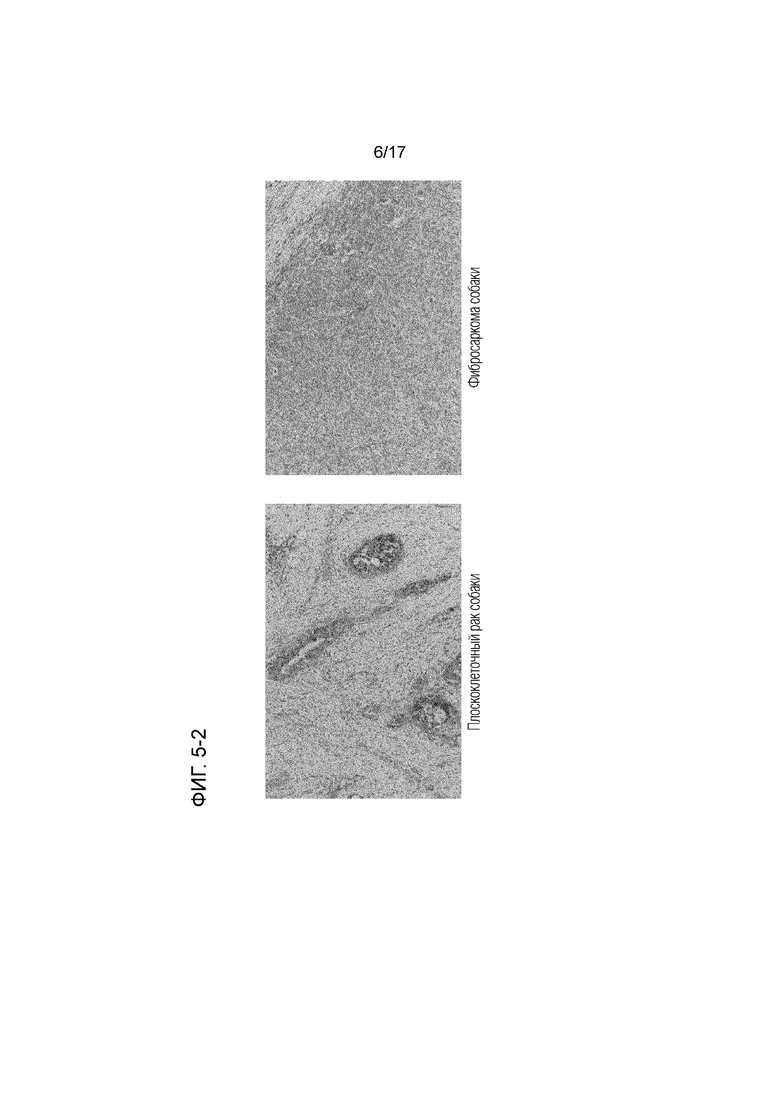




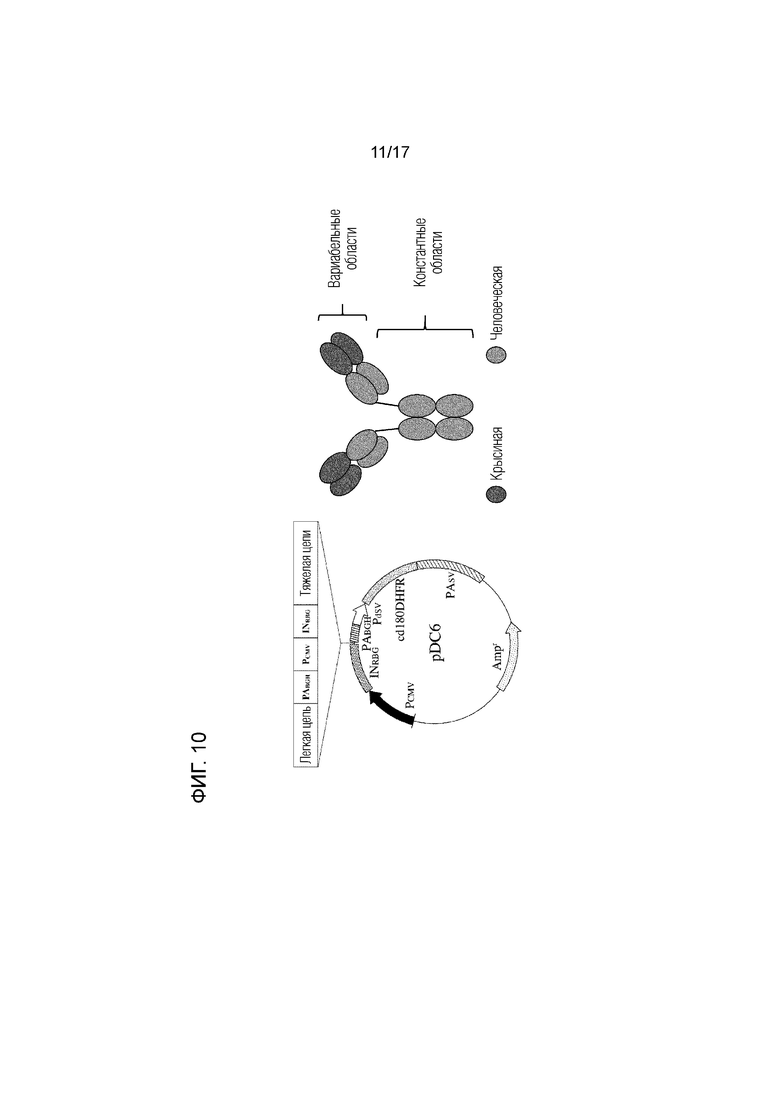




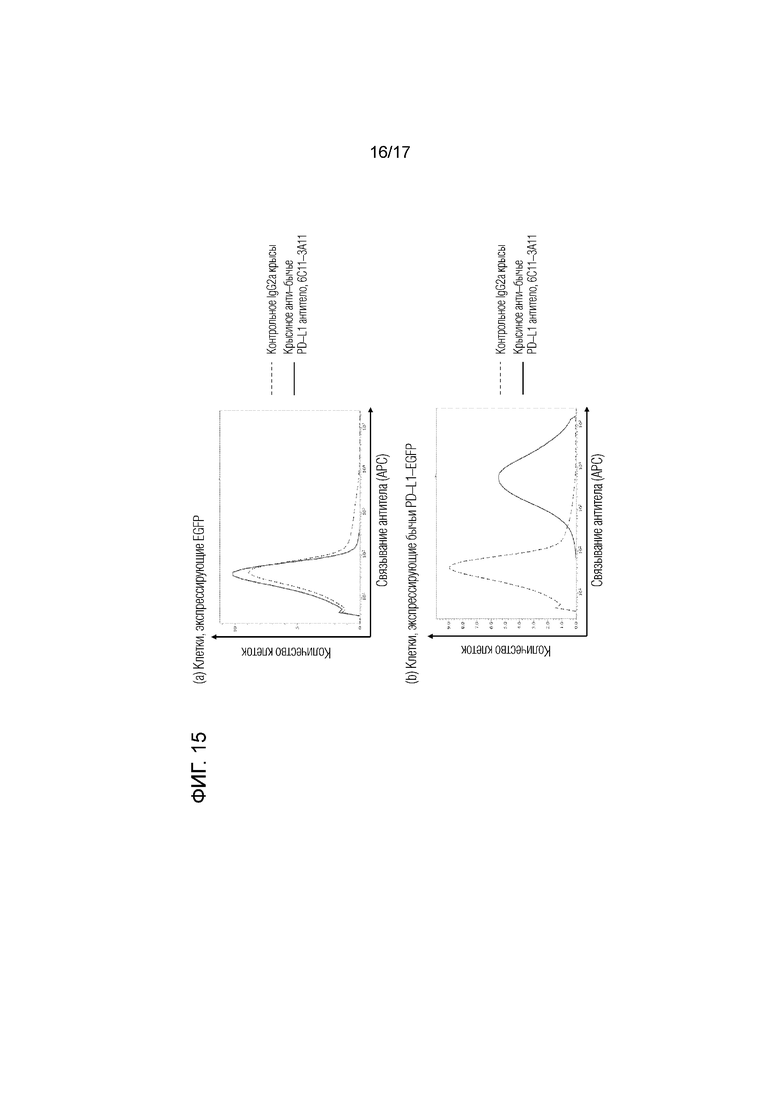

Изобретение относится к области биотехнологии. Предложено анти-PD-L1 антитело. Также предложена композиция для детекции PD–L1, содержащая вышеуказанное антитело в качестве активного ингредиента. Кроме того, изобретение направлено на кодирующие указанное антитело нуклеиновые кислоты, вектор экспрессии, клетку-хозяина и способ получения антитела. Предложенное антитело способно высокоэффективно окрашивать опухолевые клетки меланомы. 8 н. и 10 з. п. ф-лы, 17 ил., 1 табл., 5 пр.
1. Антитело против PD–L1, содержащее (а) легкую цепь, содержащую CDR1 с аминокислотной последовательностью KSISKY (SEQ ID NO:1), CDR2 с аминокислотной последовательностью SGS и CDR3 с аминокислотной последовательностью QQHNEYPLT (SEQ ID NO:2), и (b) тяжелую цепь, содержащую CDR1 с аминокислотной последовательностью GYTFTDYI (SEQ ID NO:3), CDR2 с аминокислотной последовательностью INPDSGGN (SEQ ID NO:4) и CDR3 с аминокислотной последовательностью ARGITMMVVISHWKFDF (SEQ ID NO:5).
2. Антитело по п.1, которое получено у крысы.
3. Антитело по п.2, которое представляет собой крысиное анти–бычье антитело PD–L1.
4. Антитело по п.3, где вариабельная область легкой цепи имеет аминокислотную последовательность SEQ ID NO:6 и вариабельная область тяжелой цепи имеет аминокислотную последовательность SEQ ID NO:7.
5. Антитело по любому из пп.1–4, где вариабельная область легкой цепи имеет аминокислотную последовательность вариабельной области каппа легкой цепи.
6. Антитело по любому из пп.1–5, где константная область тяжелой цепи имеет аминокислотную последовательность константной области IgG2a.
7. Антитело по п.5 или 6, где константная область легкой цепи имеет любую аминокислотную последовательность SEQ ID NO:8, 10–12 и константная область тяжелой цепи имеет любую аминокислотную последовательность SEQ ID NO:9 или 13.
8. Антитело по любому из пп.1–7, которое имеет структуру на основе четырех цепей, состоящую из двух легких цепей и двух тяжелых цепей.
9. Композиция для детекции PD–L1, содержащая антитело по любому из пп.1–8 в качестве активного ингредиента.
10. Композиция по п.9 для применения для диагностики злокачественных новообразований и/или воспалений.
11. Композиция по п.10, где злокачественные новообразования и/или воспаления выбраны из группы, состоящей из неопластических заболеваний, лейкемии, болезни Джона, анаплазмоза, бактериального мастита, микотического мастита, микоплазменных инфекций (таких как микоплазменный мастит, микоплазменная пневмония или подобных), туберкулеза, инфекции, вызыванной Theileria orientalis, криптоспоридиоза, кокцидиоза, трипаносомоза и лейшманиоза.
12. Композиция по п.9 для применения для селекции животных, подходящих для терапии анти–PD–L1 антителами.
13. ДНК, кодирующая антитело против PD–L1 по п.1.
14. Вектор экспрессии, содержащий ДНК по п.13.
15. Клетка–хозяин для получения антитела по любому из пп.1-8, где клетка трансформирована вектором по п.14.
16. Способ получения антитела, включающий культивирование клетки–хозяина по п.15 и сбор анти–PD–L1 антитела из полученной культуры.
17. ДНК, кодирующая легкую цепь антитела против PD–L1, где вариабельная область легкой цепи имеет аминокислотную последовательность SEQ ID NO:6.
18. ДНК, кодирующая тяжелую цепь антитела против PD–L1, где указанная тяжелая цепь содержит CDR1 с аминокислотной последовательностью GYTFTDYI (SEQ ID NO:3), CDR2 с аминокислотной последовательностью INPDSGGN (SEQ ID NO:4) и CDR3 с аминокислотной последовательностью ARGITMMVVISHWKFDF (SEQ ID NO:5).
| OKAGAWA TOMOHIRO et al., Bovine Immunoinhibitory Receptors Contribute to Suppression of Mycobacterium avium subsp | |||
| paratuberculosis-Specific T-Cell Responses, Infection and Immunity, January 2016, vol.84, no.1, pp.77-89 | |||
| MAEKAWA NAOYA et al., Expression of PD-L1 on Canine Tumor Cells and Enhancement of IFN-γ Production from Tumor-Infiltrating |
Авторы
Даты
2021-11-01—Публикация
2018-03-23—Подача