Область изобретения
Изобретение относится к области молекулярной диагностики, персонализированной медицины и клинической онкологии, а именно к расчету мутационной нагрузки опухоли (TMB) на миллион пар оснований с использованием данных секвенирования РНК опухоли и алгоритмов машинного обучения с учителем.
Уровень техники
В то время как глобальная угроза рака продолжает возрастать, разрабатываются новые стратегии лечения, такие как таргетная терапия и иммунотерапия. Многие из этих методов лечения полезны только для небольшого числа пациентов, что подчеркивает необходимость персонализации назначения лекарств и поиска надежных биомаркеров ответа на терапию.
Мутационная нагрузка опухоли (TMB) на миллион пар оснований является хорошо известным прогностическим маркером эффективности ингибиторов контрольных точек иммунитета (Zhu J, et al., Association Between Tumor Mutation Burden (TMB) and Outcomes of Cancer Patients Treated With PD-1/PD-L1 Inhibitions: A Meta-Analysis. Front Pharmacol. 2019;10: 673). TMB может быть определен как число соматических мутаций на миллион пар оснований (мегабаз) в белок-кодирующей области генома, включая как замены, так и делеции/инсерции, без учета функциональной роли мутаций (Chalmers ZR, et al., Analysis of 100,000 human cancer genomes reveals the landscape of tumor mutational burden. Genome Med. 2017;9: 34). Сильно мутированные опухоли с большей вероятностью продуцируют опухолевые неоантигены и являются более «видимыми» для иммунной системы, поэтому TMB считается надежной оценкой нагрузки опухоли неоантигенами (Fancello L, Gandini S, Pelicci PG, Mazzarella L. Tumor mutational burden quantification from targeted gene panels: major advancements and challenges. J Immunother Cancer. 2019;7: 183).
На сегодняшний день оценка TMB коммерчески доступна в форме диагностических тестов для клинических и исследовательских целей (Büttner R, et al. Implementing TMB measurement in clinical practice: considerations on assay requirements. ESMO Open. 2019;4: e000442). Расчёт TMB является частью многих генетических тест-системы. Все доступные в настоящее время генетические тесты используют данные секвенирования ДНК опухоли для расчета TMB. Большинство из них вычисляют TMB, используя данные таргетной панели экзомного секвенирования, реже - данные секвенирования всего экзома (WES) или секвенирования всего генома (WGS).
Профилирование экспрессии генов с использованием данных секвенирования РНК является другим типом анализа, который может помочь в назначении терапии (Buzdin A, et al. RNA sequencing for research and diagnostics in clinical oncology. Semin Cancer Biol. 2020 Feb;60:311-323). Такой тип анализа получит дополнительное преимущество, если будет включать оценку TMB. В данных по секвенированию РНК присутствуют только эскпрессируемые гены, поэтому TMB, рассчитанный по данным секвенирования РНК, потенциально может быть более надежным предиктором эффективности ингибиторов контрольных точек иммунитета, чем TMB, рассчитанный по данным WES, WGS или таргетного ДНК секвенирования панели генов.
Хотя это и возможно теоретически, но на практике надежное обнаружение соматических мутаций для расчета TMB с использованием данных секвенирования только-опухолевой РНК является проблематичным из-за ряда факторов, таких как гетерогенность опухоли, неоднородный охват ген-кодирующих областей, и артефактов, часто возникающих в результате фиксации образца опухоли. Машинное обучение (ML) в настоящее время широко используется для различных задач в биоинформатике и, в частности, для ее применения в клинической онкологии (Tkachev V, et al., Flexible Data Trimming Improves Performance of Global Machine Learning Methods in Omics-Based Personalized Oncology. Int J Mol Sci. 2020 Jan 22;21(3)). Существует необходимость в разработке проверенного алгоритма ML для улучшения расчета TMB на основе данных секвенирования только-опухолевой РНК.
Сущность изобретения
Раскрытые здесь варианты исполнения изобретения относятся к способам, системам и продуктам для расчета мутационной нагрузки опухоли (TMB) с использованием данных секвенирования только-опухолевой РНК из образцов опухоли. Цель состоит в том, чтобы разработать эффективный подход для расчета TMB на миллион пар оснований (мегабазу) на основе данных секвенирования РНК с использованием фильтрации соматических мутаций с помощью модели машинного обучения с учителем (ML). Этот подход основан на модификации стандартного набора программ для идентификации мутаций в данных секвенирования только-опухолевой РНК, с дополнительным этапом фильтрации, который выполняется моделью ML. Технический результат заключается в повышении точности расчета TMB по данным секвенирования только-опухолевой РНК.
В одном варианте исполнения предложен метод компьютерного вычисления мутационной нагрузки опухоли (TMB) с использованием данных секвенирования только-опухолевой РНК из образца опухоли, причем способ включает стадии: (а) получение данных РНК секвенирования образца опухоли, и прогнозирование мутаций в РНК с использованием набора программ для идентификации и аннотации мутаций, посредством чего создается предварительный профиль мутаций для образца опухоли; (b) применение предварительно обученной модели машинного обучения с учителем (ML) для расчета TMB образца опухоли путем корректировки предварительного профиля мутаций; где предобучение модели ML включает в себя: I. получение обучающей выборки, содержащей выборку мутаций по данным секвенирования РНК и вторую выборку мутаций из соответствующих данных секвенирования ДНК, взятых из тех же образцов опухоли; II. аннотирование мутаций в первой выборке мутаций в виде ИСТИННЫХ или ЛОЖНЫХ мутаций на основе второй выборки мутаций; III. выполнение обучения модели ML с учителем с использованием первой и второй выборок мутаций.
Несколько модификаций или альтернативных вариантов исполнения метода возможны без отклонения от сущности изобретения; некоторые из этих альтернативных вариантов реализации перечислены ниже.
В одном варианте реализации метод дополнительно содержит этап проверки модели ML после предобучения путем тестирования предобученной модели ML с учителем на второй обучающей выборке. Разделение выборок данных на обучающую выборку и тестовую выборку может быть выполнено с использованием различных порогов в соответствии с Y. Xu and R. Goodacre, J Anal Test. 2018; 2 (3): 249-262. Достаточное количество образцов должно быть оставлено в тестовой выборке для надежной оценки производительности модели путем расчета корреляции между фактическим и прогнозируемым TMB.
В другом варианте реализации образец опухоли и образцы тренировочной выборки получают из опухолевой ткани, заключенной в парафин и фиксированной в формалине (FFPE).
В другом варианте реализации образец опухоли и образцы тренировочного набора получают из свежезамороженной опухолевой ткани.
В другом варианте реализации коррекция предварительного мутационного профиля выполняется с использованием алгоритма градиентного бустинга.
В другом варианте реализации коррекция предварительного мутационного профиля выполняется с использованием алгоритма случайных деревьев.
В другом варианте реализации предобучение модели ML с учителем дополнительно включает следующие этапы: выделение признаков, которые характеризуют аннотированные мутации в первой выборке мутаций; выбор извлеченных элементов и подходящей модели для ML с учителем; классификация выбранных признаков как соответствующих ИСТИННОЙ или ЛОЖНОЙ мутации.
В другом варианте реализации предобучение модели ML с учителем выполняется до тех пор, пока метрика AUC (площадь под ROC-кривой) параметров модели не станет равной или превышающей 90% на кросс-валидации. Методы вычисления метрики AUC известны в данной области техники, см., например, Green Dm, S. J. A. (1966) Signal Detection Theory and Psychophysics, New York.
В другом варианте реализации изобретения раскрыт вычислительный продукт, причем вычислительный продукт содержит машиночитаемый носитель длительного хранения, содержащий множество инструкций для управления вычислительной системой для выполнения операции одним из вышеупомянутых способов.
В другом варианте реализации изобретения раскрыта система для расчета мутационной нагрузки опухоли (TMB) с использованием данных секвенирования только-опухолевой РНК из образца опухоли, причем система содержит: по меньшей мере, один носитель данных, сконфигурированный для хранения набора данных секвенирования, включая, по меньшей мере, данные секвенирования РНК и данные секвенирования ДНК, взятые из образцов опухоли; и, по меньшей мере, один процессор, функционально связанный с, по меньшей мере, тем одним носителем данных, причем, по меньшей мере, один процессор сконфигурирован для (а) получения данных о секвенировании РНК из образца опухоли и предсказания мутаций в РНК с помощью набора программ для идентификации и аннотирования, в результате чего создается предварительный профиль мутаций для образца опухоли; (б) применения предобученной модели машинного обучения (ML) с учителем для расчета TMB для образца опухоли путем корректировки предварительного профиля мутаций; где предварительное обучение модели ML включает в себя: I. получение обучающей выборки, содержащей первую выборку мутаций из данных секвенирования РНК, взятых из образцов опухоли, и вторую выборку мутаций из соответствующих данных секвенирования ДНК, взятых из тех же образцов опухоли; II. аннотирование мутаций в первой выборке мутаций в виде ИСТИННОЙ или ЛОЖНОЙ мутации на основе второй выборки мутаций; III. выполнение обучения модели ML с учителем с использованием первой и второй выборок мутаций.
Элементы любого из раскрытых вариантов реализации могут использоваться в сочетании друг с другом без ограничения. Кроме того, другие функции и преимущества настоящего изобретения станут очевидными для специалистов в данной области техники после рассмотрения следующего подробного описания и прилагаемых иллюстраций.
КРАТКОЕ ОПИСАНИЕ РИСУНКОВ
Подробное описание изложено со ссылкой на прилагаемые рисунки.
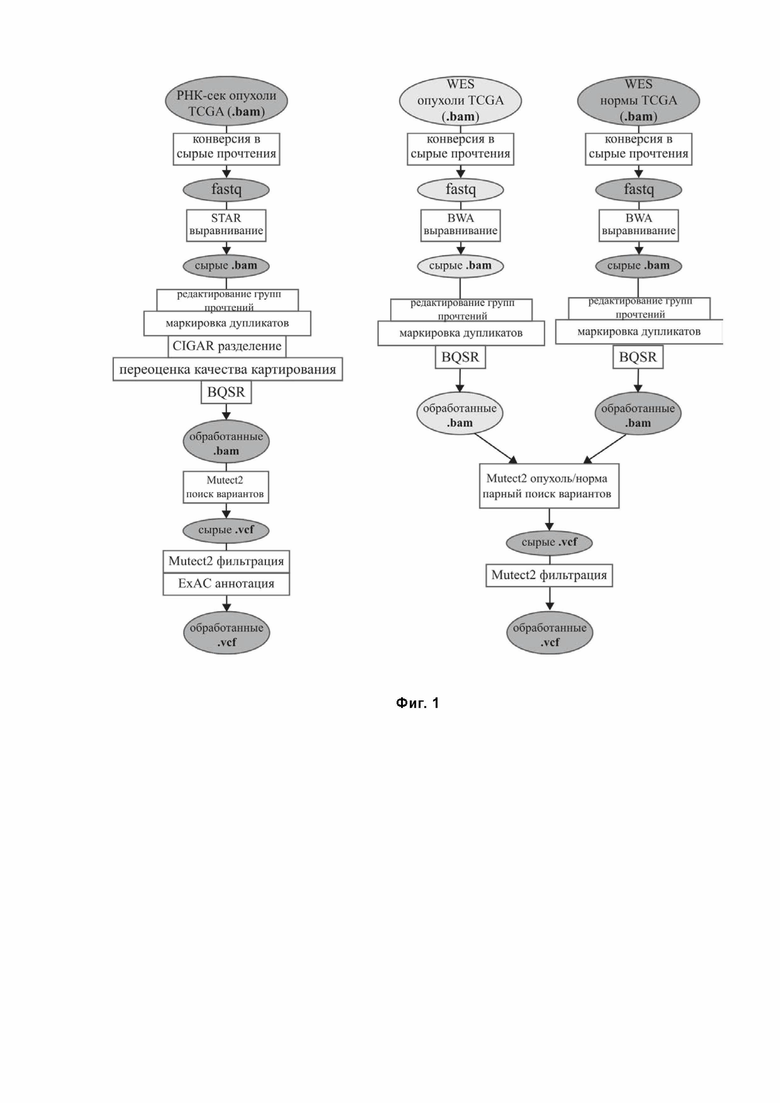
Фиг. 1. Пример набора программ для идентификации мутаций. Файлы с данными показаны в овалах, шаги набора программ - в прямоугольниках. Файлы секвенирования РНК (RNAseq) находятся на левой панели, полноэкзомного секвенирования (WES) опухолевой и WES нормальной ткани - на правой панели.
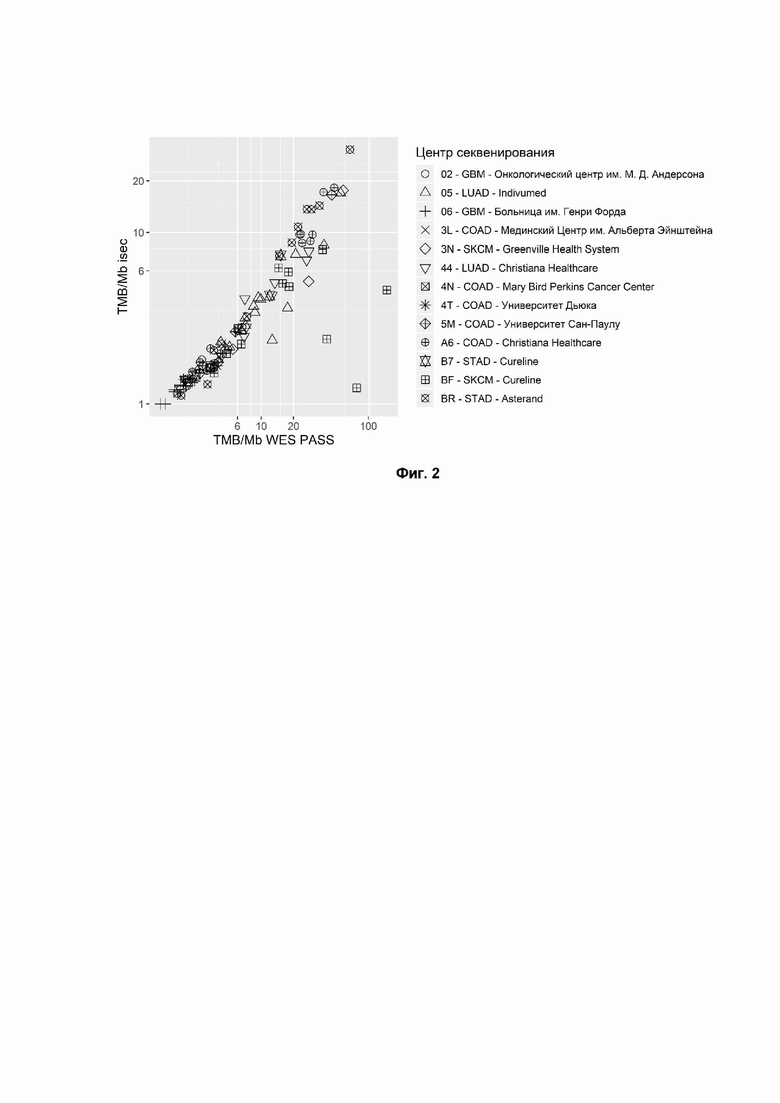
Фиг. 2. TMB, рассчитанный с использованием данных WES для регионов, также охваченных данными секвенирования РНК свежезамороженной ткани (ось Y, «isec» обозначает пересечение данных секвенирования РНК и WES) по сравнению со всем набором данных WES (ось X ). Формы точек соответствуют локализациям образцов тканей из TCGA. Коды болезни TCGA - COAD: аденокарцинома толстой кишки; GBM: мультиформная глиобластома; LUAD: аденокарцинома легкого; STAD: аденокарцинома желудка; SKCM: меланома кожи. Коэффициент корреляции Спирмена = 0,92 (р-значение < 2 *10-16), коэффициент корреляции Пирсона = 0,88 (р-значение < 2*10-16).
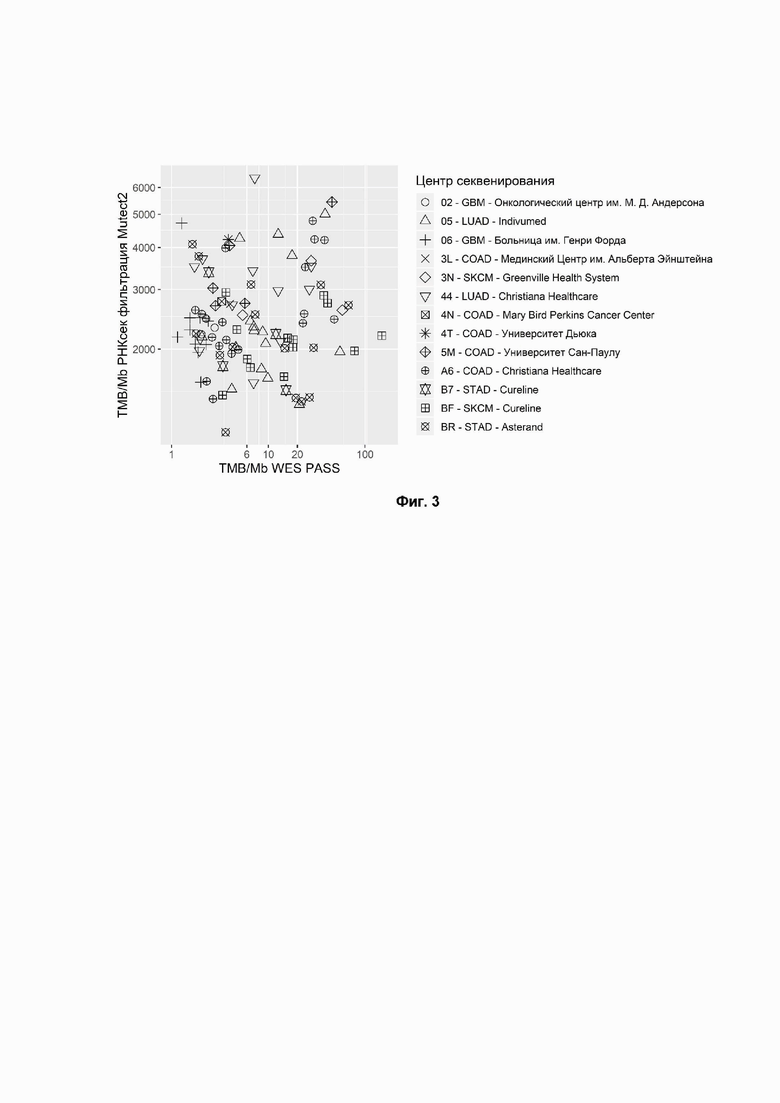
Фиг. 3. TMB, рассчитанный по данным РНК-секвенирования (RNAseq) свежезамороженной ткани, отфильтрованным с использованием стандартной фильтрации идентификатора вариантов (ось Y) по сравнению с данными WES (ось X). Формы точек соответствуют локализациям образцов тканей из TCGA. Коды болезни TCGA COAD: аденокарцинома толстой кишки; GBM: мультиформная глиобластома; LUAD: аденокарцинома легкого; STAD: аденокарцинома желудка; SKCM: меланома кожи. Коэффициент корреляции Спирмена = 0,06 (р-значение = 0,52), коэффициент корреляции Пирсона = 0,09 (р-значение = 0,359).
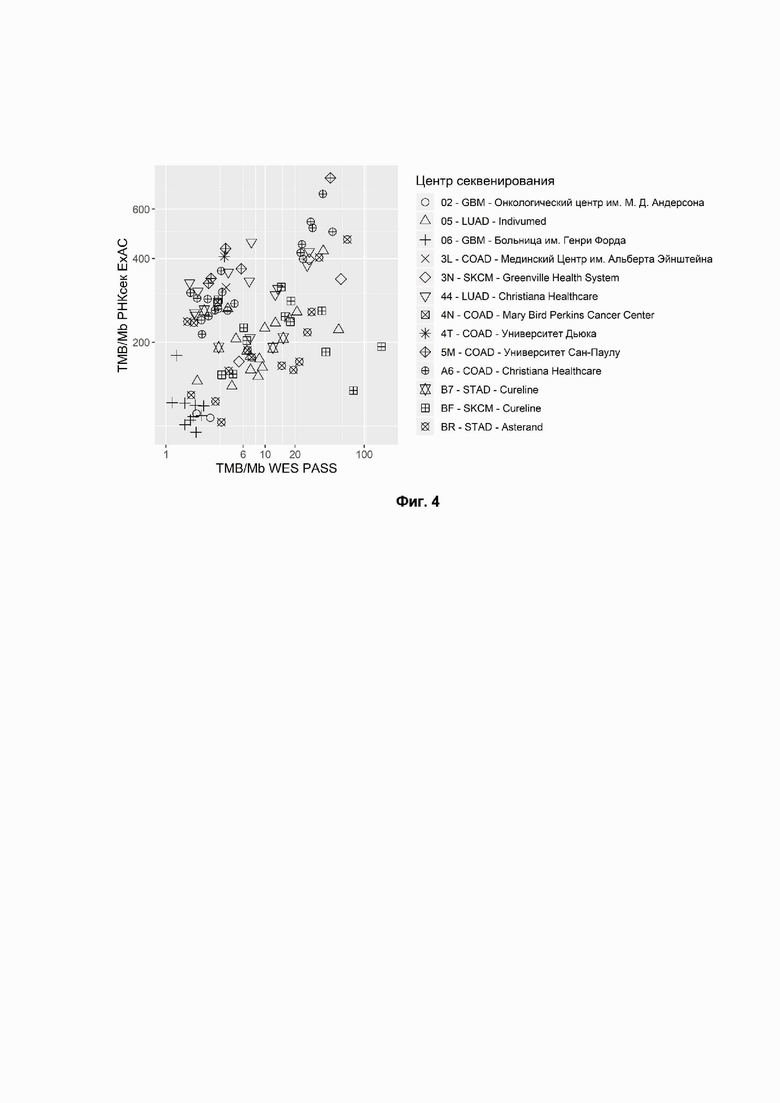
Фиг. 4. TMB, рассчитанный по данным РНК-секвенирования (RNAseq) свежезамороженной ткани, отфильтрованным с использованием стандартной фильтрации идентификатора вариантов, а также с фильтрации известных полиморфизмов из базы данных ExAC (ось Y) по сравнению с данными WES (ось X). Формы точек соответствуют локализациям образцов тканей из TCGA. Коды болезни TCGA - COAD: аденокарцинома толстой кишки; GBM: мультиформная глиобластома; LUAD: аденокарцинома легкого; STAD: аденокарцинома желудка; SKCM: меланома кожи. Коэффициент корреляции Спирмена = 0,4 (р-значение = 3.73*10-5), коэффициент корреляции Пирсона = 0,44 (р-значение = 3.09*10-6).
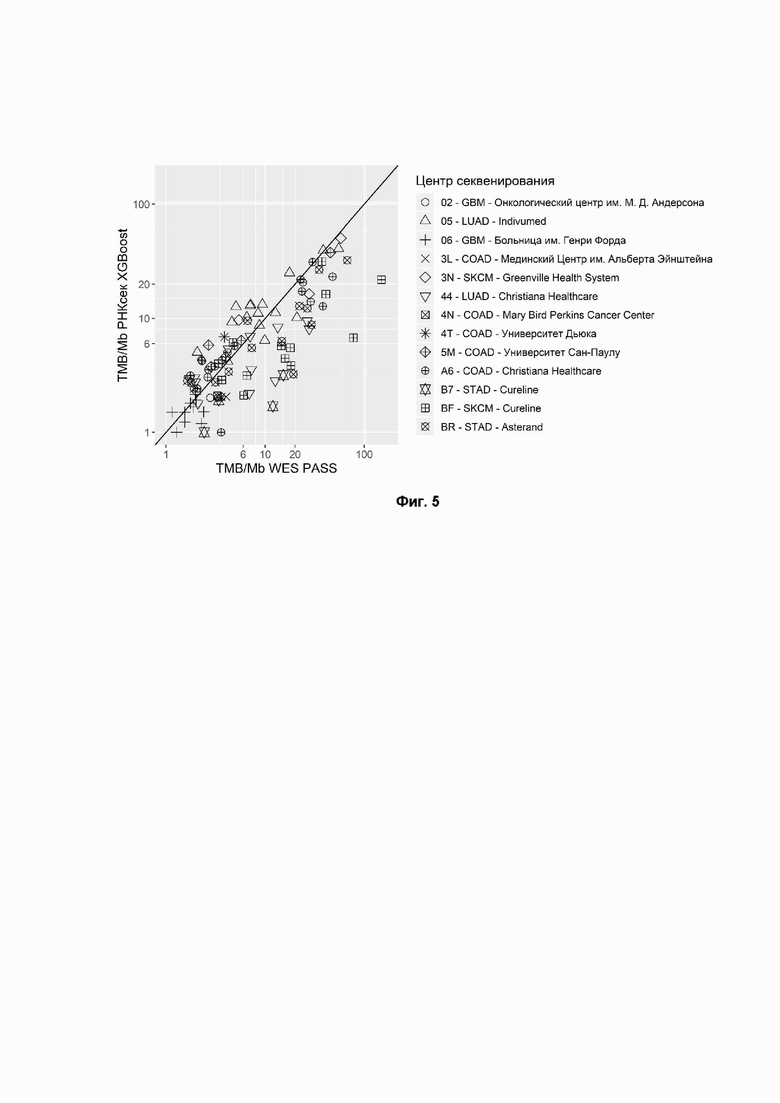
Фиг. 5. TMB, рассчитанный по данным РНК-секвенирования (RNAseq) из свежезамороженной ткани, отфильтрованным с использованием XGBoost (ось Y) по сравнению с данными WES (ось X). Формы точек соответствуют локализациям образцов тканей из TCGA. Коды болезни TCGA - COAD: аденокарцинома толстой кишки; GBM: мультиформная глиобластома; LUAD: аденокарцинома легкого; STAD: аденокарцинома желудка; SKCM: меланома кожи. Коэффициент корреляции Спирмена = 0,52 (р-значение = 0,00709), коэффициент корреляции Пирсона = 0,67 (р-значение = 0,000165).
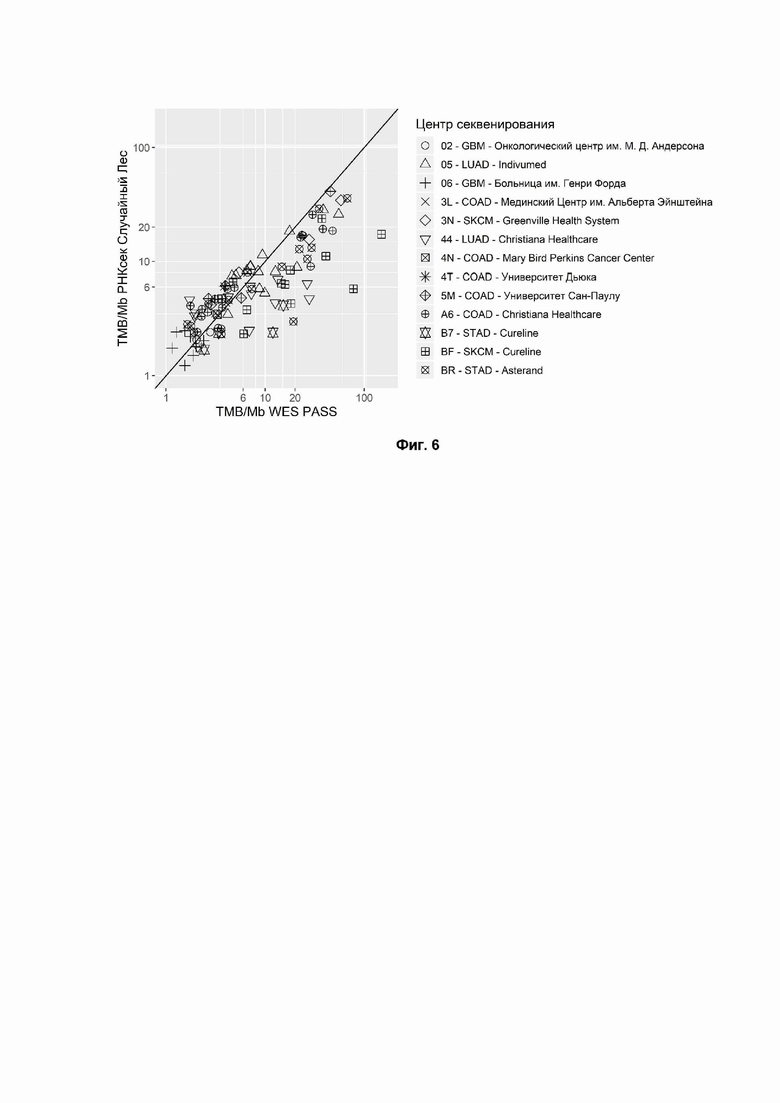
Фиг. 6. TMB, рассчитанный по данным РНК-секвенирования (RNAseq) свежезамороженной ткани, отфильтрованным с использованием метода случайных деревьев (ось Y) по сравнению с данными WES (ось X). Формы точек соответствуют локализациям образцов тканей из TCGA. Коды болезни TCGA - COAD: аденокарцинома толстой кишки; GBM: мультиформная глиобластома; LUAD: аденокарцинома легкого; STAD: аденокарцинома желудка; SKCM: меланома кожи. Коэффициент корреляции Спирмена = 0,82 (р-значение < 2*10-16), коэффициент корреляции Пирсона = 0,82 (р-значение < 2*10-16).
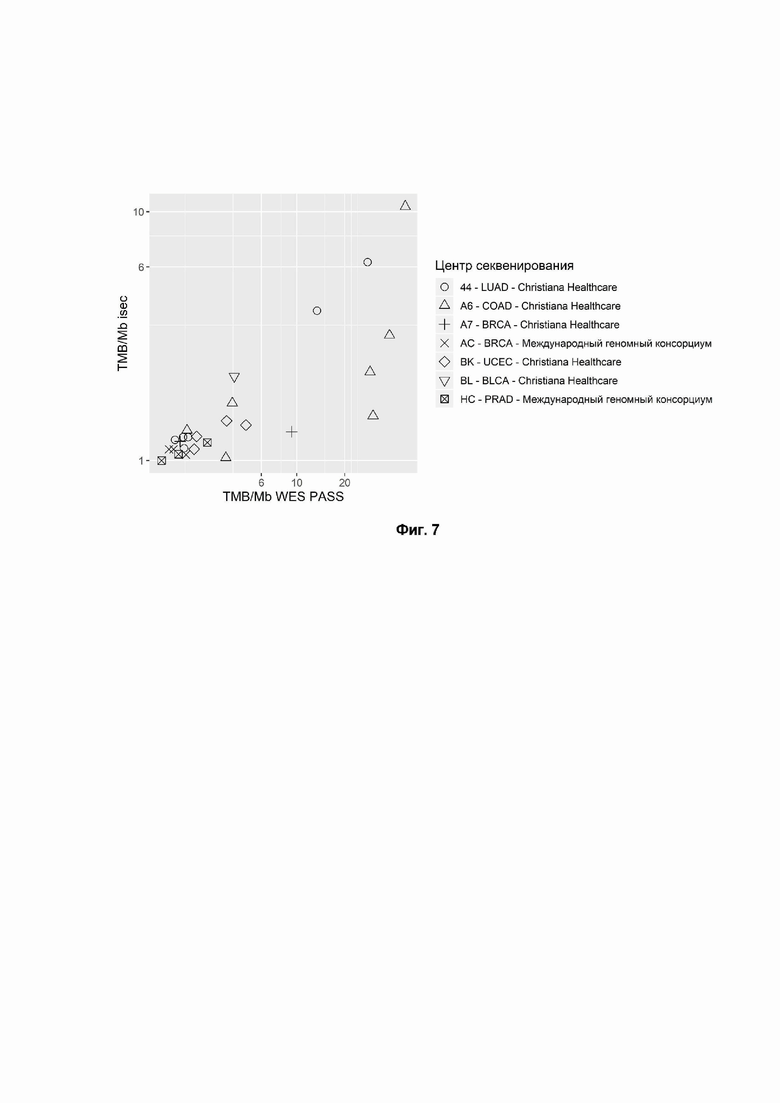
Фиг. 7. TMB, рассчитанный с использованием данных WES для регионов, также охваченных данными секвенирования РНК парафинизированной ткани (FFPE) (ось Y, «isec» обозначает пересечение данных секвенирования РНК и WES), по сравнению со всем набором данных WES (ось X). Формы точек соответствуют локализациям образцов тканей из TCGA. Коды болезни TCGA - COAD: аденокарцинома толстой кишки; LUAD: аденокарцинома легкого; BRCA: рак молочной железы; UCEC: карцинома матки и тела матки; BLCA: рак мочевого пузыря; PRAD: аденокарцинома простаты. Коэффициент корреляции Спирмена = 0,82 (р-значение = 3.28*10-7), коэффициент корреляции Пирсона = 0,82 (р-значение = 3.97*10-7).
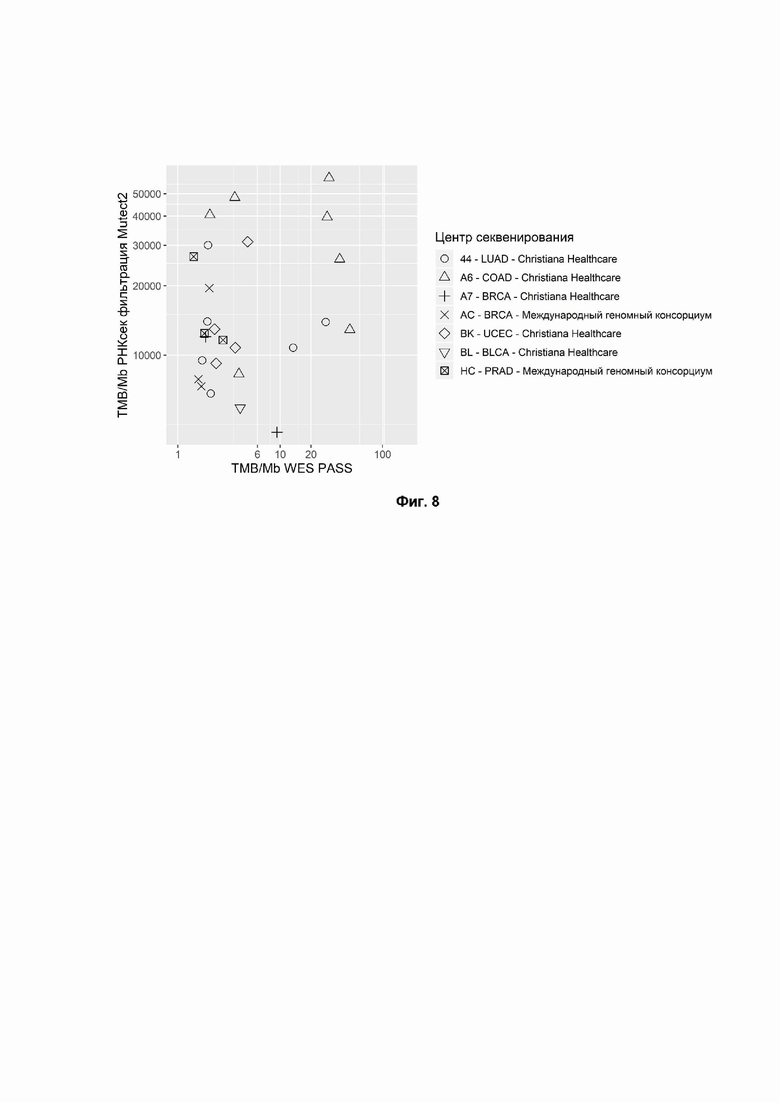
Фиг. 8. TMB, рассчитанный на основе данных секвенирования РНК парафинизированной ткани (FFPE RNAseq), отфильтрованных с использованием стандартной фильтрации идентификатора вариантов (ось Y) по сравнению с данными WES (ось X). Формы точек соответствуют локализациям образцов тканей из TCGA. Коды болезни TCGA - COAD: аденокарцинома толстой кишки; LUAD: аденокарцинома легкого; BRCA: рак молочной железы; UCEC: карцинома матки и тела матки; BLCA: рак мочевого пузыря; PRAD: аденокарцинома простаты. Коэффициент корреляции Спирмена = 0,18 (р-значение = 0,377), коэффициент корреляции Пирсона = 0,26 (р-значение = 0,207).
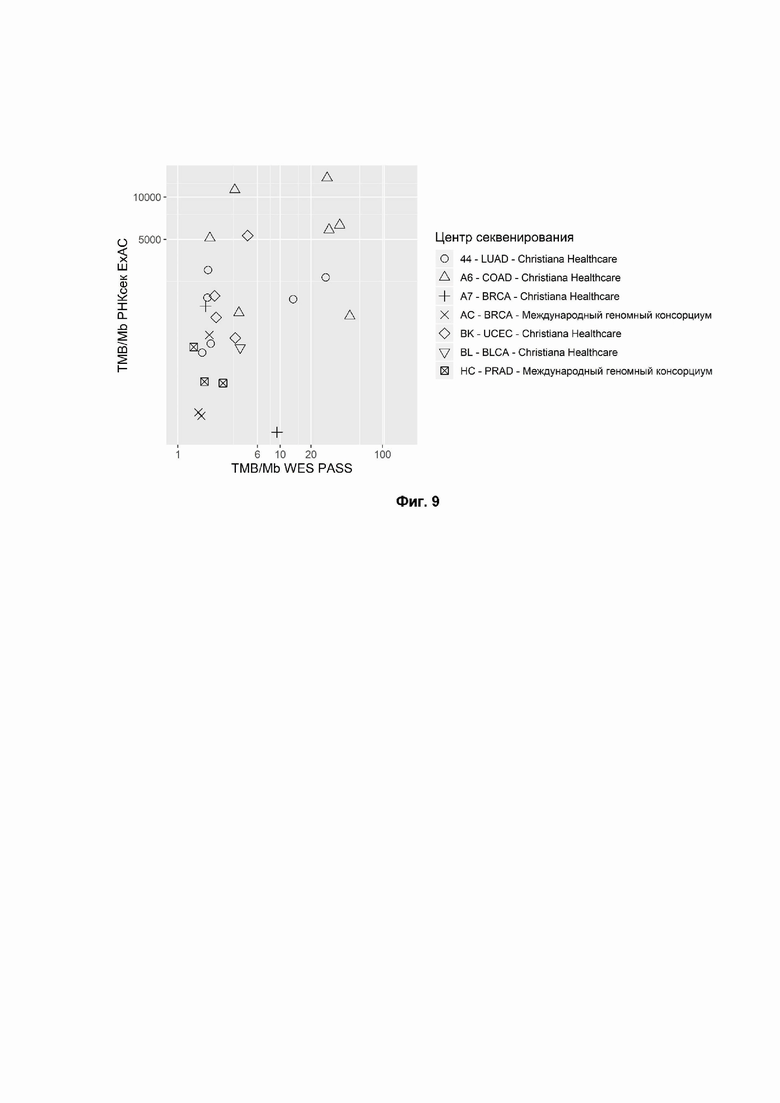
Фиг. 9. TMB, рассчитанный по данным РНК-секвенирования парафинизированной ткани (FFPE RNAseq), с использованием стандартной фильтрации идентификатора вариантов, а также фильтрации известных полиморфизмов из базы данных ExAC (ось Y) по сравнению с данными WES (ось X). Формы точек соответствуют локализациям образцов тканей из TCGA. Коды болезни TCGA - COAD: аденокарцинома толстой кишки; LUAD: аденокарцинома легкого; BRCA: рак молочной железы; UCEC: карцинома матки и тела матки; BLCA: рак мочевого пузыря; PRAD: аденокарцинома простаты. Коэффициент корреляции Спирмена = 0,49 (р-значение = 0,0122), коэффициент корреляции Пирсона = 0,45 (р-значение = 0,0211).
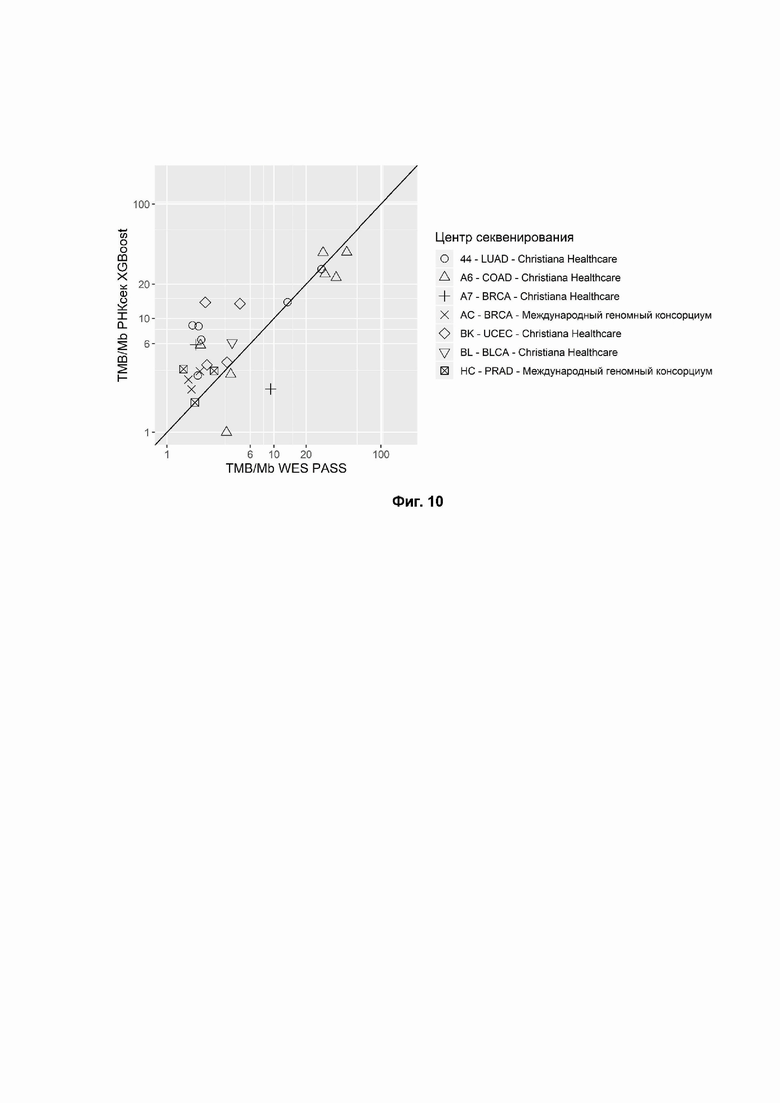
Фиг. 10. TMB, рассчитанный на основе данных секвенирования РНК парафинизированной ткани (FFPE RNAseq), отфильтрованных с использованием XGBoost (ось Y) по сравнению с данными WES (ось X). Формы точек соответствуют локализациям образцов тканей из TCGA. Коды болезни TCGA - COAD: аденокарцинома толстой кишки; LUAD: аденокарцинома легкого; BRCA: рак молочной железы; UCEC: карцинома матки и тела матки; BLCA: рак уротелия пузыря; PRAD: аденокарцинома простаты. Коэффициент корреляции Спирмена = 0,6 (р-значение = 0,00141), коэффициент корреляции Пирсона = 0,74 (р-значение = 1.68*10-5).
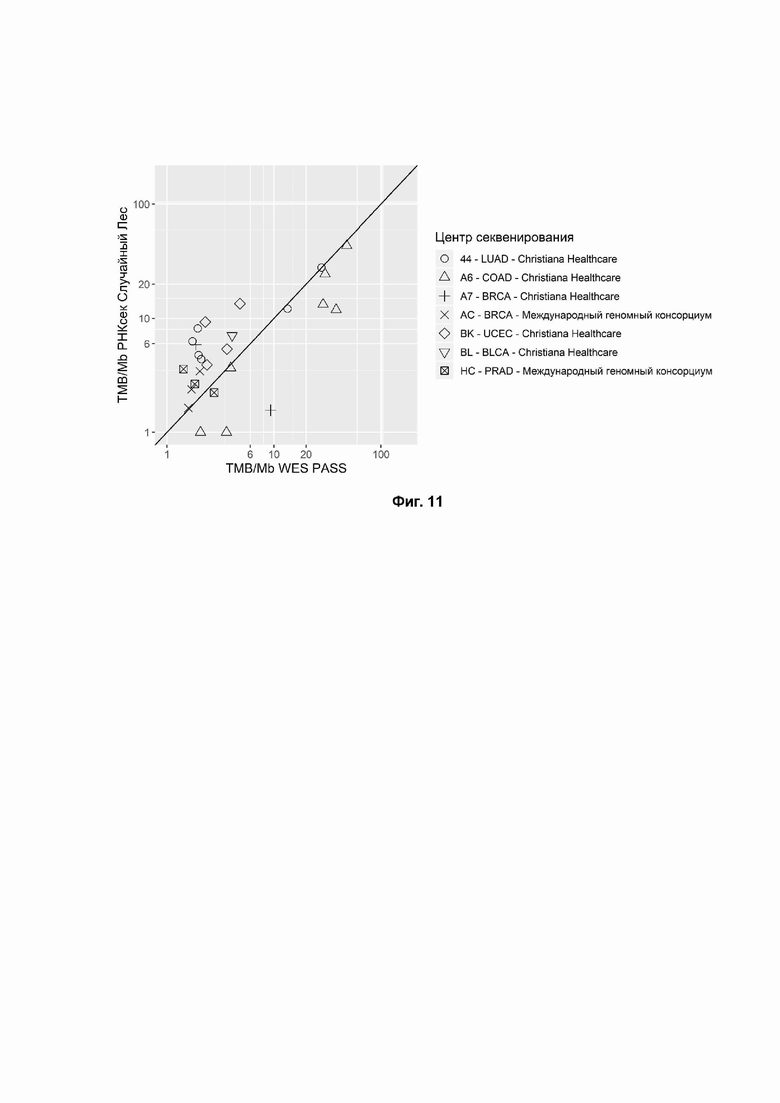
Фиг. 11. TMB, рассчитанный по данным РНК-секвенирования парафинизированной ткани (FFPE RNAseq), отфильтрованным с использованием метода случайных деревьев (ось Y) по сравнению с данными WES (ось X). Формы точек соответствуют локализациям образцов тканей из TCGA. Коды болезни TCGA - COAD: аденокарцинома толстой кишки; LUAD: аденокарцинома легкого; BRCA: рак молочной железы; UCEC: карцинома матки и тела матки; BLCA: рак мочевого пузыря; PRAD: аденокарцинома простаты. Коэффициент корреляции Спирмена = 0,58 (р-значение = 0,00202), коэффициент корреляции Пирсона = 0,69 (р-значение = 8.96*10-5).
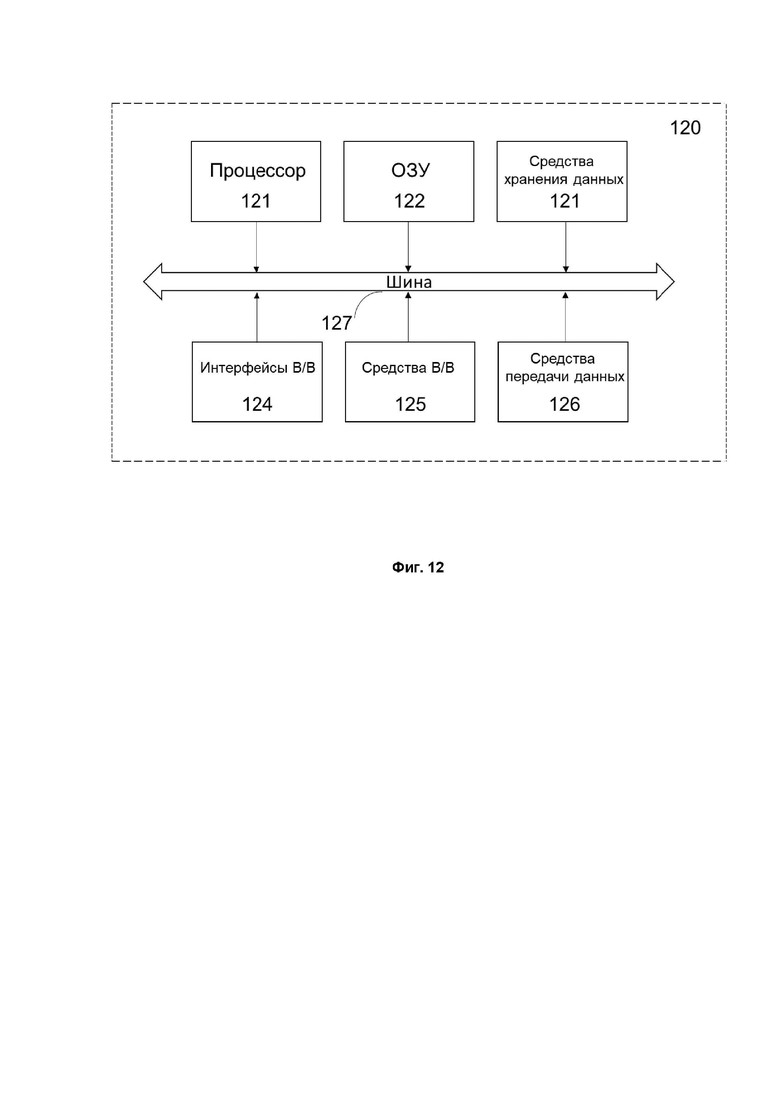
Фиг. 12. Общая схема вычислительного устройства (120), которое обеспечивает обработку данных, необходимую для реализации заявленных методов.
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
В данном описании термины «включает», «включающий» и/или «имеющий», используемые в данном документе, определяются как содержащий (то есть открытый язык). Эти термины не предназначены для толкования как «состоит только из». Термин «другой», используемый в данном документе, определяется как, по меньшей мере, второй или более. Термин «множество», используемый в данном документе, определяется как два или более двух. Упоминание на протяжении всего этого документа слов как «один вариант реализации», «определенные варианты реализации», «вариант реализации», «реализация», «пример» или других подобных терминов означает, что конкретная особенность, структура или характеристика, описанная в связи с вариантом реализации, включена, по меньшей мере, в одном варианте реализации настоящего изобретения. Таким образом, появление таких фраз в различных местах данного описания не обязательно относится к одному и тому же варианту реализации. Кроме того, конкретные особенности, структуры или характеристики могут быть объединены любым подходящим способом в одном или нескольких вариантах реализации без ограничения. Технические и научные термины в данном описании имеют стандартные значения, общепринятые в научно-технической литературе, если не указано иное.
Соматическая мутация - это генетическая вариация, которая произошла во время онкогенеза. Такие мутации обнаруживаются в опухолях, но отсутствуют в здоровой ткани того же человека.
Мутации зародышевой линии - это вариации, которые были унаследованы человеком от его/ее родителей или произошли в гаметах или на ранних стадиях эмбрионального развития.
«Модель машинного обучения с учителем» означает задачу машинного обучения, состоящую в обучении функции, которая отображает входные данные в выходные на основе примеров пар ввода-вывода из маркированных данных обучающей выборки.
«Мутационная нагрузка опухоли на мегабазу (TMB)» определяется как число соматических мутаций на мегабазу анализируемой кодирующей области, включая однонуклеотидные замены, вставки, делеции, без учета функциональных эффектов мутаций.
В некоторых вариантах реализации изобретения обработка данных секвенирования РНК с помощью набора программ для идентификации соматических мутаций в режиме анализа опухолевого образца, включает такие этапы, как: 1) выравнивание прочтений секвенирования с референсным геномом или транскриптомом, 2) использование набора программ для идентификации мутаций без учета нормальной ткани, с или без панели нормальных (здоровых) тканей.
В предпочтительных вариантах реализации изобретения, данные секвенирования РНК из образца опухоли анализируются без какого-либо сопоставленного образца нормальной (здоровой) ткани, например, полученного из крови или прилегающих к опухоли нормальных тканей от того же человека.
Для создания предварительного профиля мутаций для опухолевого образца, в предпочтительных вариантах реализации изобретения получают данные секвенирования РНК из образца опухоли и прогнозируют мутационную нагрузку опухоли при помощи алгоритмического поиска и аннотирования мутаций. Под алгоритмическим поиском следует понимать использование стандартных, специализированных программных инструментов, известных специалистам. Некоторые из них раскрыты ниже, например, GATK-подобный пайплайн идентификации мутаций.
«Панель данных нормальной ткани» означает набор отсеквенированных образцов здоровой ткани от группы индивидуумов. Этот набор предназначен быть репрезентативным для вариантов зародышевой линии, распространенных в популяции в целом, а также используется для обнаружения технических артефактов с целью улучшения результатов идентификации мутаций.
«Соответствующий образец здоровой ткани» означает образец здоровой ткани того же индивидуума, включая, но не ограничивая, кровь или здоровую ткань, прилегающую к опухоли.
Для машинного обучения с учителем необходима выборка данных для обучения. В некоторых вариантах реализации изобретения выборка обучающих данных содержит образцы секвенирования РНК опухоли с аннотированными мутациями. Аннотация подразумевает исследование статуса наличия мутации в опухоли и соответствующих образцов здоровой ткани в отдельном эксперименте или серии экспериментов для валидации истинных соматических мутаций. Если мутация обнаружена в опухоли и не обнаружена в нормальном образце, такой вариант аннотируется как истинная соматическая мутация. Если статус мутации не может быть оценен, например, из-за низкого покрытия секвенирования, такая мутация должна быть исключена из обучающей выборки данных.
В настоящем описании «предобучение» модели ML означает выполнение тренировки модели ML перед фактическим использованием, чтобы скорректировать предварительный профиль мутаций для вычисления TMB для образца опухоли.
«Отдельный эксперимент» означает любой эксперимент или серию экспериментов, необходимых для определения мутационного статуса (соматической или зародышевой линии) в опухоли. Такие эксперименты сравнивают данные, полученные из опухоли, с соответствующими образцами здоровой ткани и могут включать, но не ограничиваются, секвенирование всего генома или экзома, панель таргентного секвенирования ДНК и обнаружение полиморфизмов с помощью микрочиповой гибридизации ДНК.
Характеристики мутаций, идентифицированных соответствующим набором программ для идентификации соматических мутаций, которые будут использоваться для обучения с учителем, могут быть получены из параметров прочтений, параметров выравнивания, контекста мутации и положения в геноме, а также из внешних аннотаций, таких как базы данных известных полиморфизмов и функциональной аннотации мутаций и т.д.
Секвенирование РНК и ДНК может быть выполнено для нуклеиновых кислот, выделенных из тканей человека, хранящихся в различных условиях: свежезамороженных тканей, заархивированных тканей, фиксированных формалином и заключенных в парафиновые блоки (FFPE), тканей, хранящихся в RNAlater или другом стабилизирующем растворе, примером чего являются следующие научные публикации (Suntsova M. et al., Atlas of RNA sequencing profiles for normal human tissues. Sci Data. 2019 Apr 23;6(1):36; Hedegaard J. et al. Next-generation sequencing of RNA and DNA isolated from paired fresh-frozen and formalin-fixed paraffin-embedded samples of human cancer and normal tissue. PLoS One. 2014 May 30;9(5):e98187; Choi Y. et al. Optimization of RNA Extraction from Formalin-Fixed Paraffin-Embedded Blocks for Targeted Next-Generation Sequencing. J Breast Cancer. 2017 Dec;20(4):393-399). Эти способы включены в описание изобретения.
В некоторых вариантах реализации изобретения параметры алгоритма обучения с учителем могут быть установлены путем перекрестной валидации, например, 5× или 10×. После этого выполняется подбор параметров модели на тренировочной выборке данных.
В предпочтительном варианте реализации отдельная аннотированная выборка образцов, которая не используется при первоначальном обучении (или подборе) модели, может быть сохранена в качестве дополнительной тестовой выборки для установления эффективности фильтрации. Подходящая и проверенная модель может быть использована для расчета TMB в образцах опухоли от онкопациентов.
Известно, что разные модели ML будут основывать свои прогнозы на характеристиках (признаках) мутаций совершенно по-разному и нет единых решений. Таким образом, предварительное обучение модели ML с учителем может включать различные этапы. Например, в случае линейной регрессии можно выяснить, какие признаки принимаются моделью как важные - и модели будут присваивать более высокие по абсолютной величине коэффициенты таким признакам. Для нейронных сетей очень трудно определить важность каждой характеристики. Существуют специальные методы для объяснения поведения обученной нейронной сети. Как правило, для проведения предобучения требуется только набор данных для секвенирования РНК с правильно помеченными состояниями мутации (такие как, ИСТИННАЯ или ЛОЖНАЯ), и тогда каждая модель ML будет выявлять важные для классификации признаки в своем собственном стиле. В некоторых вариантах реализации изобретения для всех категориальных признаков мутации (когда переменная принимает не числовые значения, а, например, типы мутаций A-> T, G-> A и т.д.), метод one hot encoding используется для кодирования функций.
В некоторых вариантах реализации изобретения перекрестная проверка параметров модели ML может быть выполнена следующим образом. Для выбора гиперпараметров модели ML используется классический метод разделения известного маркированного набора данных на три выборки: обучающую, тестовую и проверочную. Меньшая часть обычно откладывается для использования в качестве проверочной выборки. Большая часть делится на k частей, обычно 5 или 10, в зависимости от объема данных. Затем данные для обучения генерируются различными способами из k-1 таких частей, а оставшаяся k-я часть преобразуется в тестовую выборку. Используя этот метод кросс-валидации, можно определить точные гиперпараметры. На последнем этапе выбранная модель проверяется с использованием проверочной выборки.
В некоторых вариантах реализации изобретения предлагаются способы расчета мутационной нагрузки опухоли на мегабазу (TMB) в образцах секвенирования РНК. Подход основан на модификации стандартного набора программ для идентификации мутаций на данных секвенирования только-опухолевой РНК с дополнительным этапом фильтрации после идентификации. Фильтрация выполняется с помощью ML модели, обученной на данных секвенирования РНК, в которых наличие соматических мутаций установлено с использованием альтернативной технологии, включающей исследование статуса мутаций в опухоли и здоровых тканях. Чтобы полностью понять, как модель ML может быть обучена и реализована, эта заявка будет описывать изобретение в контексте использования модели XGBoost (T. Chen, C. Guestrin. XGBoost: a scalable tree boosting system. ArXiv, 1603 (2016). arXiv:1603.02754) и модели случайных деревьев (A. Liaw and M. Wiener (2002). Classification and Regression by randomForest. R News 2(3), 18-22) для фильтрации мутаций RNAseq. Примеры расчета TMB приведены ниже для раскрытия характеристик данного изобретения, и их никоим образом не следует рассматривать как ограничивающие объем изобретения. Следует понимать, что другие модели ML могут быть успешно обучены и использованы в различных вариантах реализации изобретения.
Примерами таких моделей ML являются линейная регрессия, логистическая регрессия, нейронные сети (Перцептрон, Многослойный перцептрон (MLP), метод обратного распространения ошибки, стохастический градиентный спуск, сеть Хопфилда, сеть радиально-базисных функций (RBFN)), алгоритмы глубокого обучения (свёрточные нейронные сети (CNN), рекуррентные нейронные сети (RNNs), сети долгой краткосрочной памяти (LSTMs), авто-энкодеры, машина Больцмана (DBM), глубокие сети доверия (DBN)), методы решающих деревьев (деревья классификации и доверия (CART), итеративный дихотомайзер 3 (ID3), C4.5 и C5.0 (различные его версии), Хи-квадратичная автоматическая детекция взаимодействий (CHAID), Decision Stump, модель M5, условные решающие деревья), метод опорных векторов (SVM), сети векторного квантования, обучаемые с учителем (LVQ), самоорганизующаяся карта Кохонена (SOM), локально взвешенное обучение (LWL), метод случайного леса, Гауссовские процессы, стохастический градиентный спуск, метод оценивания коэффициентов линейной регрессионной модели (LASSO), эластичная сеть с наименьшим углом регрессии (LARS), ridge регрессия, Баесовские алгоритмы (наивный байес, гауссовский наивный байес, мультиномиальный наивный байес, усредненные оценщики с одной зависимостью (AODE), байесовские сети доверия (BBN), байесовская сеть (BN)), метод k-ближайших соседей, алгоритмы понижения размерности (метод главных компонент (PCA), регрессия главных компонент (PCR), регрессия частично наименьших квадратов (PLSR), Sammon Mapping, метод многомерного шкалирования (MDS), поиска наилучшей проекции, линейных дискриминантный анализ (LDA), дискриминантный анализ смеси (MDA), квадратичный дискриминантный анализ (QDA), гибкий дискриминантный анализ (FDA)), градиентный бустинг (например, GBM, XGBoost, LightGBM, CatBoost), ансамбльные методы (e.g. AdaBoost, Weighted Average, Bootstrapped Aggregation) и другие.
Пример 1. Расчет TMB в 119 образцах секвенирования РНК.
Для расчета TMB с использованием данных РНК-секвенирования (RNAseq) и сравнения их с данными WES, 119 случаев пяти различных типов рака (аденокарцинома толстой кишки, мультиформная глиобластома, аденокарцинома легкого, меланома кожи и аденокарцинома желудка) из базы данных The Cancer Genome Atlas (TCGA) были проанализированы. TCGA - это база генетических данных онкопациентов, в которой содержатся результаты молекулярного профилирования более 20 000 онкопациентов и соответствующих нормальных образцов, охватывающая 33 типа рака (Cancer Genome Atlas Research Network, Weinstein JN, Collisson EA, et al., The Cancer Genome Atlas Pan-Cancer analysis project. Nat Genet. 2013 Oct;45(10):1113-20.). Данные WES и RNAseq для каждого случая идентифицированных мутаций были проанализированы, как описано ниже. 19 образцов были использованы для обучения модели ML, в то время как 100 были использованы для проверки модели.
Таблица 1. Свежезамороженные биологические образцы TCGA, использованные для обучения и проверки модели XGBoost для фильтрации мутаций RNAseq.
Образцы данных были загружены с портала GDC (https://portal.gdc.cancer.gov/). Все полученные файлы bam были конвертированы в формат fastq с помощью инструммента Picard v2.18.17 SamToFastq (http://broadinstitute.github.io/picard), с последующим выравниванием и идентификацией мутаций.
Для данных RNAseq использовался GATK-подобный пайплайн идентификации мутаций (Фиг. 1). GATK относится к Genome Analysis ToolKit (Van der Auwera GA, Carneiro MO, et al., From FastQ data to high confidence variant calls: the Genome Analysis Toolkit best practices pipeline. Curr Protoc Bioinformatics. 2013;43:11.10.1-11.10.33), разработан организацией Broad Institute (broadinstitute.org) и содержит несколько модулей для обработки данных NGS. Прочтения были выравнены на 38 версию генома человека с помощью программного обеспечения STAR v2.6.1d в режиме 2-PASS (Dobin et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 2013 Jan; 29(1): 15-21). Параметрами не по умолчанию были: sjdbOverhang 100, twopass1readsN 10000000, twopassMode Basic. Координаты экзонов были взяты из аннотации Ensembl версии 89. Samtools v1.3.1 использовался для индексации bam (Li H., Handsaker B., Wysoker A., et al., 1000 Genome Project Data Processing Subgroup. The Sequence alignment/map (SAM) format and SAMtools. Bioinformatics. 2009 Aug 15;25(16):2078-9). Редактирование информации о группе прочтений (все прочтения были присвоены к одной группе) и маркировка дубликатов выполнялась с помощью Picard (http://broadinstitute.github.io/picard) AddOrReplaceReadGroups и MarkDuplicatesRead, соответственно. GATK v3.8.0 SplitNCigarReads модуль использовался для отделения прочтений, которые выравнивались на регионы альтернативного сплайсинга, а ReassignOneMappingQuality использовался для обеспечения совместимости качества выравнивания STAR с последующим анализом. Повторная калибровка показателя качества оснований была выполнена с помощью модулей GATK v4.beta.1 BaseRecalibrator и ApplyBQSR. Для идентификации мутации был использован GATK4 Mutect2 (Cibulskis K, Lawrence MS, et al. Sensitive detection of somatic point mutations in impure and heterogeneous cancer samples. Nat Biotechnol. 2013 Mar;31(3):213-9) в только-опухолевом (tumor-only) режиме с базой данных генетических вариантов dbSNP версии 146 (Sherry ST, Ward MH, et al., dbSNP: the NCBI database of genetic variation. Nucleic Acids Res. 2001 Jan 1;29(1):308-11) и базой данных Mills, содержащей стандартные инсерции и делеции найденные в рамках проекта 1000 геномов (Mills RE, Luttig CT, et al., An initial map of insertion and deletion (INDEL) variation in the human genome. Genome Res. 2006 Sep;16(9):1182-90). Панель норм не использовалась. Варианты идентифицировались только в экзонах (по аннотации GENCODE (Frankish A, Diekhans M, et al. GENCODE reference annotation for the human and mouse genomes. Nucleic Acids Res. 2019 Jan 8;47(D1):D766-D773), только хромосомы 1-22, X и Y) и параметр PCR_indel_model был установлен на «HOSTILE». Варианты были отфильтрованы с помощью GATK4 FilterMutectCalls (все варианты хранятся в VCF, редактируется только поле «FILTER»). Влияние мутаций на последовательность белка было аннотировано с использованием программного обеспечения annovar (Wang K, Li M, Hakonarson H. ANNOVAR: functional annotation of genetic variants from high-throughput sequencing data. Nucleic Acids Res. 2010 Sep;38(16):e164). Для ExAC (Lek M. et al. Analysis of protein-coding genetic variation in 60,706 humans. Nature. 2016 Aug 18;536(7616):285-91) аннотации, была использована часть, не содержащая нормальных тканей TCGA, и, следовательно, не включающая варианты зародышевой линии, обнаруженные у пациентов из TCGA. Все три- или более аллельных сайтов были исключены из дальнейшего анализа.
Для анализа данных WES использовался GATK-подобный пайплайн идентификации соматических мутаций (Фиг.1). Чтения были картированы на 38 версию генома человека с помощью программного обеспечения BWA mem v0.7.17. (Li H, Durbin R. Fast and accurate long-read alignment with Burrows-Wheeler transform. Bioinformatics. 2010 Mar 1;26(5):589-95). Параметры не по умолчанию были: -k 15, -r 2. Преобразование файлов sam в bam и сортировка файлов bam выполнялись с помощью программного обеспечения samtools (Li H., Handsaker B., Wysoker A., Fennell T., Ruan J., Homer N., Marth G., Abecasis G., Durbin R. and 1000 Genome Project Data Processing Subgroup. The Sequence alignment/map (SAM) format and SAMtools. Bioinformatics. 2009 Aug 15;25(16):2078-9). Остальные этапы предварительной обработки были идентичны шагам для данных RNAseq, за исключением пропущенных шагов редактирования качества прочтения и картрирования. Для идентификации мутаций, GATK4 Mutect2 (Cibulskis K, Lawrence MS et al. Sensitive detection of somatic point mutations in impure and heterogeneous cancer samples. Nat Biotechnol. 2013 Mar;31(3):213-9) использовали одновременно для опухолевых и соответствующих нормальных образцов с использованием тех же баз данных dbSNP и инсерций, делеций, что и для RNAseq. Последующие этапы постобработки включали фильтрацию GATK4 FilterMutectCalls и аннотацию annovar. Все три- или более аллельных сайтов были исключены из дальнейшего анализа. Для управления параллельными вычислительными задачами использовалось программное обеспечение GNU parallel (Tange, O. GNU Parallel-the command-line power tool. USENIX. 2011; 36, 42-47).
Для обработки данных ДНК и РНК могут использоваться и другие наборы программ для идентификации мутаций, например: FreeBayes (E. Garrison, Marth G. Haplotype-based variant detection from short-read sequencing (2012) arXiv preprint arXiv:12073907), SAMtools (H. Li. A statistical framework for SNP calling, mutation discovery, association mapping and population genetical parameter estimation from sequencing data. Bioinformatics, 27 (21) (2011), pp. 2987-2993), Platypus (A. Rimmer, H. Phan, I. Mathieson, Z. Iqbal, S.R. Twigg, A.O. Wilkie, et al., Integrating mapping-, assembly-and haplotype-based approaches for calling variants in clinical sequencing applications Nat Genet, 46 (8) (2014), pp. 912-918), SNVSniffer (Y. Liu, M. Loewer, S. Aluru, Schmidt B. SNVSniffer: an integrated caller for germline and somatic single-nucleotide and indel mutations BMC Syst Biol, 10 (2) (2016), p. 47), VarScan2 (D.C. Koboldt, Q. Zhang, D.E. Larson, D. Shen, M.D. McLellan, L. Lin, et al. Varscan 2: somatic mutation and copy number alteration discovery in cancer by exome sequencing. Genome Res, 22 (3) (2012), pp. 568-576) и другие, например, представленные в (C. Xu. A review of somatic single nucleotide variant calling algorithms for next-generation sequencing data. Comput Struct Biotechnol J. 2018 Feb 6;16:15-24).
Для ML фильтрации были выбраны 32 признака, такие как покрытие референсного аллеля, медианное качество оснований или число событий в гаплотипе. Из них 24 параметра были взяты непосредственно из выходного файла VCF Mutect2, а одна была получена из аннотации VCF с базой данных ExAC. Другие 7 параметров были построены с использованием данных из выходного VCF Mutect2: 4 логических параметра, указывающих является ли мутация (1) инсерцией, (2) делецией, (3) заменой C-> T (G-> A), (4) заменой C-> A (G-> T) и 3 численных параметра: (5) покрытие, и (6-7) длина инсерции/делеции.
Обучающий набор данных использовался для обучения модели XGBoost (T. Chen, C. Guestrin. XGBoost: a scalable tree boosting system. ArXiv, 1603 (2016). arXiv:1603.02754). Гиперпараметры модели были выбраны во время серии рандомизированных поисков по сетке. Были установлены следующие параметры: learning_rate, n_estimators, min_child_weight, gamma, subsample, colsample_bytree, max_depth, reg_alpha, reg_lambda; среди них первые два имели наибольший вес. Кросс-валидация была 5-кратной, и ROC AUC использовался в качестве метрики для выбора гиперпараметров.
Набор обучающих данных также использовался для обучения модели случайного леса (A. Liaw and M. Wiener (2002). Classification and Regression by randomForest. R News 2(3), 18-22) с 20 деревьями. Могут быть использованы другие модели машинного обучения с учителем (ML).
Во-первых, были сравнены TMB в WES и TMB в регионах WES, также покрытых в RNAseq (Фиг. 2). Это было сделано для оценки максимально возможной прогностической эффективности RNAseq. TMB, рассчитанный с использованием мутаций на основе WES в областях, покрытых в RNAseq, сильно коррелирует с TMB, рассчитанным для полного набора данных WES (Коэффициент корреляции Пирсона=0.88, P-значение<2e-16).
Эффективность фильтрации данных RNAseq оценивалась с использованием программного обеспечения Mutect2 с настройками по умолчанию (Фиг. 3, Коэффициент корреляции Пирсона=0.09, P-значение=0.359) и настройками по умолчанию в сочетании с фильтрацией вариантов зародышевой линии из не-TCGA подмножества базы данных ExAC (Фиг. 4, Коэффициент корреляции Пирсона=0.5, P-значение=3.09e-6). Была рассчитана корреляция TMB (RNAseq с фильтрацией XGBoost) с TMB (WES) (Фиг. 5, Коэффициент корреляции Пирсона=0.67, P-значение=0.000165). Также была вычислена корреляция TMB (RNAseq с фильтрацией методом случайного леса) с TMB (WES) (Фиг. 6, Коэффициент корреляции Пирсона=0.82, P-значение=2e-16). Оказалось, что фильтрация с помощью ML модели значительно превосходила наивную (стандартную) фильтрацию в оценке TMB.
Кроме того, было проверена прогностическая эффективность TMB на основе RNAseq с использованием расчета площади под ROC кривой (AUC) в тех же группах сравнения, как описано выше. Для этого использовали наиболее распространенные пороги для данных WES (TMB >6, >10 и >20, соответсвенно (Stenzinger A. et al. Tumor mutational burden standardization initiatives: Recommendations for consistent tumor mutational burden assessment in clinical samples to guide immunotherapy treatment decisions. 2019 Aug;58(8):578-588) и вычислили, является ли TMB из соответствующего профиля RNAseq предиктором TMB по WES. Было обнаружено, что подход, основанный на ML, превосходил фильтрацию по ExAC и наивную фильтрацию и может использоваться для надежной оценки TMB для всех вышеупомянутых порогов с AUC> 0,86 (таблица 2).
Таблица 2. Значения ROC AUC для прогнозирования TMB (пороговые значения: > 6,> 10 и > 20) в WES с использованием данных RNAseq.
Пример 2. Расчет TMB по данным секвенирования РНК в 50 образцах, фиксированных в формалине и заключенных в парафиновые блоки (FFPE).
Чтобы рассчитать TMB с использованием данных РНК-секвенирования (RNAseq) и сравнить его с данными WES, были проанализированы 50 пациентов восьми различных типов рака (рак мочевого пузыря, инвазивный рак молочной железы, плоскоклеточный рак шейки матки и аденокарцинома шейки матки, аденокарцинома толстой кишки, почечно-клеточный рак, аденокарцинома легкого, аденокарцинома предстательной железы и рак эндометрия матки) с данными RNAseq, доступными для фиксированных в формалине и заключенных в парафиновые блоки (FFPE) образцов в базе данных The Cancer Genome Atlas (TCGA) (Таблица 3).
TCGA - это база генетических данных онкопациентов, в которой содержатся результаты молекулярного профилирования более 20 000 онкопациентов и соответствующих нормальных образцов, охватывающая 33 типа рака (Cancer Genome Atlas Research Network, Weinstein JN, Collisson EA, et al., The Cancer Genome Atlas Pan-Cancer analysis project. Nat Genet. 2013 Oct;45(10):1113-20). Данные WES и RNAseq для каждого случая идентифицированных соматических мутаций для 27 образцов были использованы для обучения модели ML, в то время как 23 были использованы для проверки модели.
Таблица 3. Биологические образцы TCGA FFPE, используемые для обучения и проверки модели XGBoost для фильтрации мутаций, идентифицированных из данных RNAseq.
Анализ RNAseq и WES, а также ML фильтрация были проведены аналогично Примеру 1.
В случае образцов FFPE TMB, рассчитанный с использованием мутаций на основе WES в областях, покрытых RNAseq, сильно коррелирует с TMB, рассчитанным для полного набора данных WES (Фиг. 7, коэффициент корреляции Пирсона = 0,82, P-значение = 3,97e-7).
Эффективность фильтрации данных RNAseq оценивалась с использованием программного обеспечения Mutect2 с настройками по умолчанию. (Cibulskis K, Lawrence MS, Carter SL, Sivachenko A, Jaffe D, Sougnez C, et al. Sensitive detection of somatic point mutations in impure and heterogeneous cancer samples. Nat Biotechnol. 2013 Mar;31(3):213-9) (Фиг. 8, коэффициент корреляции Пирсона = 0.26, P-значение=0.207) и с настройками по умолчанию в сочетании с фильтрацией вариантов зародышевой линии из не-TCGA подмножества ExAC (Lek M. et al. Analysis of protein-coding genetic variation in 60,706 humans. Nature. 2016 Aug 18;536(7616):285-91) (Фиг. 9, Коэффициент корреляции Пирсона=0.45, P-значение=0.0211). Была посчитана корреляция TMB (RNAseq с XGBoost фильтрацией) с TMB (WES) (Фиг. 10, Коэффициент корреляции Пирсона=0.74, P-значение=1.68e-5). Так же была вычислена корреляция TMB (RNAseq с фильтрацией по методу случайных деревьев) с TMB (WES) (Фиг. 11, Коэффициент корреляции Пирсона=0.69, P-значение=8.96e-5). Фильтрация с помощью ML-модели значительно превзошла наивную (стандартную) фильтрацию в оценке TMB.
Прогнозирующая эффективность RNAseq TMB затем была протестирована с использованием AUC ROC анализа в тех же группах сравнения, как описано выше. Были использованы те же пороговые значения, что и в примере 1 для данных WES (TMB >6, >10 и >20), чтобы оценить, является ли TMB из соответствующего профиля RNAseq предиктором TMB из WES. ML подход превзошел ExAC и наивную фильтрацию в большинстве случаев и мог быть использован для надежной оценки TMB в образцах FFPE для всех вышеуказанных порогов с AUC ≥ 0,85 (Таблица 4).
Таблица 4. Значения ROC AUC для прогнозирования TMB (пороговые значения: > 6,> 10 и > 20) в WES с использованием данных FFPE RNAseq.
На Фиг. 12 представлена общая схема вычислительного устройства (120), которое обеспечивает обработку данных, необходимую для реализации заявленных методов. Обычно устройство (120) содержит следующие компоненты: один или несколько процессоров (121), по меньшей мере, одну память (122), по меньшей мере, один носитель данных (123), интерфейсы ввода/вывода (I/O) (124), средства для ввода-вывода данных (125), сетевые инструменты (126). Процессор (121) выполняет основные вычислительные операции, необходимые для работы устройства (120) или функциональности одного или нескольких его компонентов. Процессор (121) выполняет необходимые машиночитаемые инструкции, расположенные в оперативной памяти (122). Память (122) выполнена обычно в виде оперативной памяти и содержит необходимую программную логику, обеспечивающую необходимые функциональные возможности. Носитель данных (123) представлен в виде жестких дисков или дисков SSD, массивов, сетевых хранилищ, флэш-памяти, оптических устройств хранения информации (CD, DVD, MD, Blue-Ray) и т.д. Носитель данных (123) обеспечивает долговременное хранение различных типов информации, например, вышеупомянутых данных РНК и ДНК-секвенирования, алгоритмов ML, идентификаторов пользователей и т.д.
Интерфейсы ввода/вывода (124) обычно являются стандартными инструментами для подключения и работы со стороны сервера, например, USB, RS232, RJ45, LPT, COM, HDMI, PS/2, Lightning, FireWire и т.д. На выбор I/O интерфейсы (124) зависят от специфики устройства (120), которым может быть персональный компьютер, мэйнфрейм, кластер серверов, смартфон, ноутбук и т.д.
В качестве средства для данных ввода/вывода (125) в любом варианте реализации системы, которая использует описанные способы, предпочтительно использовать клавиатуру. Аппаратная версия клавиатуры может быть любой известной: это может быть встроенная клавиатура, используемая на ноутбуке или нетбуке, или автономное устройство, подключенное к настольному компьютеру, серверу или другому вычислительному устройству. Соединение может быть проводным, в котором соединительный кабель клавиатуры подключен к порту PS/2 или USB, расположенному на системном блоке настольного компьютера, или беспроводным, в котором клавиатура обменивается данными, например, по беспроводному каналу; радиоканал с базовой станцией, которая, в свою очередь, напрямую подключена к системному блоку, например, к одному из портов USB. Помимо клавиатуры, данные ввода/вывода также могут включать в себя: джойстик, дисплей (сенсорный экран), проектор, сенсорную панель, мышь, трекбол, световое перо, динамики, микрофон и т.д.
Инструменты сетевой связи (126) или сетевые инструменты включают в себя устройство, которое обеспечивает сетевой прием и передачу данных, например, карту Ethernet, модуль WLAN/Wi-Fi, модуль Bluetooth, модуль BLE, модуль NFC, IrDa, модуль RFID, GSM-модем и т.д. С помощью сетевых инструментов (126) обеспечивается организация обмена данными по проводному или беспроводному каналу данных, например, WAN, PAN, LAN, LAN, Интранет, Интернет, WLAN, WMAN или GSM.
Наконец, компоненты устройства (120) обычно соединяются через общую шину данных (127).
Вышеприведенное описание раскрывает и описывает только примерные варианты реализации настоящего изобретения. Как будет понятно специалистам в данной области техники, настоящее изобретение может быть воплощено в других конкретных формах без отклонения от его сущности или существенных характеристик. Многочисленные модификации и вариации настоящего изобретения возможны в свете вышеизложенного. Соответственно, раскрытие настоящего изобретения предназначено для иллюстрации, но не ограничения объема изобретения, который изложен в следующей формуле изобретения. Раскрытие, включая любые легко различимые варианты приведенных здесь идей, частично определяет объем терминологии формулы изобретения, так что ни один предмет изобретения не предназначен для широкой публики.
Все публикации, заявки на патенты, патенты и другие ссылки, упомянутые здесь, включены в качестве ссылки во всей их полноте. Кроме того, материалы, способы и примеры являются только иллюстративными и не предназначены для ограничения, если не указано иное.
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ прогнозирования выживаемости у пациентов с аденокарциномой легкого, несущей мутации в гене KRAS | 2019 |
|
RU2702986C1 |
| Компьютерно-реализуемый интегральный способ для оценки качества результатов таргетного секвенирования | 2018 |
|
RU2717809C1 |
| СПОСОБЫ ВЫЯВЛЕНИЯ РАКА ЛЕГКОГО | 2017 |
|
RU2760913C2 |
| СПОСОБ ДИФФЕРЕНЦИАЛЬНОЙ ДИАГНОСТИКИ ТИПА РАКА ЖЕЛУДКА МЕТОДОМ ИММУНОГИСТОХИМИЧЕСКОГО ТЕСТИРОВАНИЯ | 2023 |
|
RU2813670C1 |
| СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ ОБНАРУЖЕНИЯ СОМАТИЧЕСКОГО ВАРИАНТА | 2019 |
|
RU2813655C2 |
| МЕТОД | 2016 |
|
RU2739036C2 |
| ИДЕНТИФИКАЦИЯ, ПРОИЗВОДСТВО И ПРИМЕНЕНИЕ НЕОАНТИГЕНОВ | 2016 |
|
RU2729116C2 |
| СПОСОБЫ И СИСТЕМЫ МОНИТОРИНГА СОСТОЯНИЯ ЗДОРОВЬЯ И ПАТОЛОГИИ ОРГАНОВ | 2020 |
|
RU2818052C2 |
| ПИРИДИНОВЫЕ СОЕДИНЕНИЯ ПЛАДИЕНОЛИДА И СПОСОБЫ ПРИМЕНЕНИЯ | 2015 |
|
RU2807278C2 |
| СПОСОБЫ ЛЕЧЕНИЯ РАКА, ИМЕЮЩЕГО ГЕМИЗИГОТНУЮ ПОТЕРЮ ТР53 | 2016 |
|
RU2721953C2 |
Группа изобретений относится к медицине, а именно к области молекулярной диагностики, персонализированной медицины и клинической онкологии. Предложен способ вычисления мутационной нагрузки опухоли (TMB) на миллион пар оснований на основании данных секвенирования РНК из образца опухоли в качестве альтернативы для секвенирования полного экзома или таргетной панели генов. Фильтрация мутаций по данным секвенирования РНК, используемая в наборах программ для идентификации мутаций, дополнена фильтрацией после идентификации и аннотирования мутаций с использованием машинного обучения с истинным набором мутаций, полученным из опухолей и соответствующих нормальных тканей, проверенных методом секвенирования полного экзома. Группа изобретений обеспечивает увеличение коэффициента корреляции для TMB, измеренного с помощью РНК секвенирования, увеличение коэффициента корреляции между TMB, измеренным двумя различными методами, с 0,18 до 0,6. 3 н. и 11 з.п. ф-лы, 4 табл., 12 ил., 2 пр.
1. Реализуемый на компьютере способ расчета мутационной нагрузки опухоли (ТМВ) с использованием данных секвенирования РНК из образца опухоли, включающий следующие этапы:
(a) получают данные секвенирования РНК из образца опухоли и прогнозируют мутационную нагрузку опухоли при помощи алгоритмического поиска и аннотирования мутаций, посредством чего создается предварительный профиль мутаций для опухолевого образца;
(b) применяют предварительно обученную модель машинного обучения с учителем (ML) для расчета ТМВ для образца опухоли путем корректировки предварительного профиля мутаций,
где предварительное обучение модели ML включает:
i. получение обучающей выборки, содержащей первый набор мутаций из данных секвенирования РНК, взятых из образцов опухоли, и второй набор мутаций по данным секвенирования ДНК, взятых соответственно из одних и тех же образцов опухолей;
ii. аннотирование мутаций в первом наборе мутаций в виде ИСТИННЫХ или ЛОЖНЫХ мутаций на основе второго набора мутаций;
iii. выполнение обучения модели ML с использованием первого и второго наборов мутаций.
2. Способ по п. 1, дополнительно содержащий этап проверки модели ML после предварительной тренировки путем тестирования предварительно обученной модели ML на втором тренировочном наборе.
3. Способ по п. 1, в котором образец опухоли и образцы из обучающего набора получают из опухолевой ткани, фиксированной в формалине и заключенной в парафин.
4. Способ по п. 1, в котором образец опухоли и образцы из тренировочного набора получают из свежезамороженной опухолевой ткани.
5. Способ по п. 1, в котором коррекцию предварительного профиля мутаций выполняют с использованием алгоритма градиентного бустинга.
6. Способ по п. 1, в котором исправление предварительного профиля мутации выполняется с использованием алгоритма случайного леса.
7. Способ по п. 1, в котором предварительное обучение модели ML дополнительно содержит следующие этапы:
извлечение признаков, которые характеризуют аннотированные мутации в первом наборе мутаций;
выбор извлеченных признаков и обучение модели ML;
классификация выбранных признаков как соответствующих ИСТИННОЙ или ЛОЖНОЙ мутации.
8. Способ по п. 1, в котором предварительное обучение модели ML выполняется до тех пор, пока показатель AUC по кросс-валидации параметров модели не станет равным или превышающим 90%.
9. Предназначенный для долговременного хранения информации машиночитаемый носитель, хранящий машиночитаемые указания, которые, будучи исполненными системой, включающей по меньшей мере одно вычислительное устройство, инициируют реализацию способа расчета мутационной нагрузки опухоли (ТМВ) с использованием данных секвенирования РНК из образца опухоли по п. 1.
10. Система для расчета мутационной нагрузки опухоли (ТМВ) с использованием данных секвенирования РНК из образца опухоли, включающая:
по меньшей мере, один носитель данных, сконфигурированный для хранения множества данных секвенирования, включая, по меньшей мере, данные секвенирования РНК и данные секвенирования ДНК, взятые из образцов опухоли; а также по меньшей мере, один процессор, функционально связанный с, по меньшей мере, одним носителем данных, причем, по меньшей мере, один процессор сконфигурирован с целью:
(a) получения данных секвенирования РНК из образца опухоли и прогнозирования мутационной нагрузки опухоли при помощи алгоритмического поиска и аннотирования мутаций, создавая тем самым предварительный профиль мутаций для опухолевого образца;
(b) применения предварительно обученной модели машинного обучения с учителем (ML) для расчета ТМВ для образца опухоли путем корректировки предварительного профиля мутаций,
где предварительное обучение модели ML включает:
i. получение обучающей выборки, содержащей первый набор мутаций из данных секвенирования РНК, взятых из образцов опухоли, и второй набор мутаций от данных секвенирования ДНК, взятых соответственно из одних и тех же образцов опухолей;
ii. аннотирование мутаций в первом наборе мутаций в виде ИСТИННЫХ или ЛОЖНЫХ мутаций на основе второго набора мутаций;
iii. выполнение обучения модели ML с использованием первого и второго наборов мутаций.
11. Система по п. 10, в которой предварительное обучение модели ML дополнительно содержит этапы:
извлечение признаков, которые характеризуют аннотированные мутации в первом наборе мутаций;
выбор извлеченных признаков и обучение ML модели;
классификация выбранных признаков как соответствующих ИСТИННОЙ или ЛОЖНОЙ мутации.
12. Система по п. 10, в которой исправление предварительного профиля мутаций выполняется с использованием алгоритма градиентного бустинга.
13. Система по п. 10, в которой исправление предварительного профиля мутаций выполняется с использованием алгоритма случайного леса.
14. Система по п. 10, в которой предварительное обучение модели ML выполняется до тех пор, пока показатель AUC по кросс-валидации параметров модели не станет равным или превышающим 90%.
| Zhu J., et al., Association Between Tumor Mutation Burden (TMB) and Outcomes of Cancer Patients Treated With PD-1/PD-L1 Inhibitions: A Meta-Analysis | |||
| Front Pharmacol | |||
| Станок для придания концам круглых радиаторных трубок шестигранного сечения | 1924 |
|
SU2019A1 |
| WO 2014149134 A2, 25.05.2014 | |||
| Büttner R., et al., Implementing TMB measurement in clinical practice: considerations on assay requirements | |||
| ESMO Open | |||
| Станок для придания концам круглых радиаторных трубок шестигранного сечения | 1924 |
|
SU2019A1 |
Авторы
Даты
2021-11-10—Публикация
2020-08-31—Подача