ПЕРЕКРЕСТНАЯ ССЫЛКА НА РОДСТВЕННЫЕ ЗАЯВКИ
[0001] Данная заявка заявляет преимущество и приоритет по предварительной заявке США 62/268333, поданной 16 декабря 2015 г., предварительной заявке США 62/317823, поданной 4 апреля 2016 г., предварительной заявке США 62/379986, поданной 26 августа 2016 г., предварительной заявке США 62/394074, поданной 13 сентября 2016 года, и предварительной заявке США 62/425995, поданной 23 ноября 2016 года, каждая из которых включена в качестве ссылки во полном объеме для всех целей.
УРОВЕНЬ ТЕХНИКИ
[0002] Терапевтические вакцины, основанные на опухолеспецифических неоантигенах, имеют большие перспективы в качестве нового поколения персонализированной иммунотерапии рака.1-3 Виды рака с высокой мутационной нагрузкой, такие как немелкоклеточный рак легкого (НМРЛ) и меланома, являются особенно привлекательными мишенями такой терапии, учитывая относительно большую вероятность возникновения неоантигена.4,5 Ранние данные свидетельствуют о том, что вакцинация на основе неоантигенов может вызывать Т-клеточные реакции6 и что клеточная терапия, нацеленная на неоантиген, может приводить к регрессии опухоли при определенных обстоятельствах у отдельных пациентов.7
[0003] Один из вопросов при проектировании неоантигенной вакцины заключается в том, какая из многих кодирующих мутаций, присутствующих в опухолях субъекта, может генерировать «лучшие» терапевтические неоантигены, например, антигены, которые могут вызывать противоопухолевый иммунитет и приводить к регрессии опухоли.
[0004] Были предложены первичные методы, включающие анализ на основе мутаций с использованием секвенирования нового поколения, экспрессию гена(ов) на уровне РНК и прогнозирование аффинности связывания ГКГС пептидов кандидатных неоантигенов8. Однако эти предлагаемые способы не могут моделировать весь процесс создания эпитопа, который включает много этапов (например, ТАР транспорт (ТАР - транспортер, ассоциированный с процессингом антигенов), протеасомальное расщепление и/или TCR распознавание (TCR - Т-клеточный рецептор) в дополнение к экспрессии гена(ов) и связыванию ГКГС9. Следовательно, существующие способы, вероятно, будут иметь значительно уменьшенную прогностическую ценность положительного результата (PPV). (Фиг. 1А)
[0005] Действительно, анализ пептидов, презентированных опухолевыми клетками, проведенными несколькими группами, показал, что <5% пептидов, которые, как прогнозируется, будут презентированы с использованием экспрессии генов и аффинности связывания ГКГС, могут быть обнаружены на ГКГС опухолевой поверхности10,11 (фиг. 1В). Эта низкая корреляция между прогнозированием связывания и ГКГС презентацией была еще более усилена недавними наблюдениями за отсутствием повышения точности прогнозирования рестриктированных по связыванию неоантигенов для ответа ингибитора контрольной точки по сравнению с количеством только мутаций.12
[0006] Эта низкая прогностическая ценность положительного результата (PPV) существующих способов прогнозирования презентации представляет собой проблему для разработки вакцины на основе неоантигена. Если вакцины разрабатываются с использованием прогнозов с низким PPV, большинство пациентов вряд ли получат терапевтический неоантиген, и менее вероятно, что они получат более одного (даже если все презентированные пептиды являются иммуногенными). Таким образом, неоантигенная вакцинация существующими методами вряд ли будет успешной для значительного числа субъектов, имеющих опухоль. (Фиг. 1С)
[0007] Кроме того, предыдущие подходы приводили к созданию кандидатных неоантигенов с использованием только действующих в цис-положениях мутаций и в основном пренебрегали рассмотрением дополнительных источников нео-ОРС, включая мутации в факторах сплайсинга, которые встречаются во многих типах опухолей и приводят к аберрантному сплайсированию многих генов13, и мутации, которые создают или удаляют сайты расщепления протеазы.
[0008] Наконец, стандартные подходы к анализу опухолевого генома и транскриптома могут пропустить соматические мутации, которые приводят к появлению потенциальных неоантигенов из-за субоптимальных состояний в конструировании библиотеки, экзомном и транскриптомном захвате, секвенировании или анализе данных. Аналогичным образом, стандартные подходы к анализу опухолей могут непреднамеренно вызывать артефакты последовательностей или полиморфизмы зародышевых линий в качестве неоантигенов, что приводит к неэффективному использованию потенциала вакцины или риску аутоиммунных реакций, соответственно.
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
[0009] В данном документе раскрыт оптимизированный подход для идентификации и отбора неоантигенов для персонализированных противораковых вакцин. Во-первых, рассматриваются оптимизированные подходы к анализу опухолевой экзомы и транскриптомы для идентификации неоантигенных кандидатов с использованием секвенирования нового поколения (NGS). Данные способы основаны на стандартных подходах к анализу опухолей при помощи NGS, чтобы удостовериться в том, что высокая чувствительность и специфичность неоантигенных кандидатов улучшаются для всех классов геномных изменений. Во-вторых, предлагаются новые подходы к выбору неоантигена с высоким PPV для преодоления проблемы специфичности и обеспечения того, что неоантигены, улучшенные по включению вакцины, более склонны вызывать противоопухолевый иммунитет. Эти подходы включают, в зависимости от варианта осуществления, обученные модели статистической регрессии или нелинейные модели глубокого обучения, которые совместно моделируют сопоставления аллель-пептид, а также мотивов на аллель для пептида с разными длинами, используя статистическую мощность между пептидами разной длины. Нелинейные модели глубокого обучения, в частности, могут быть разработаны и обучены для обработки различных аллелей ГКГС в одной и той же ячейке независимо друг от друга, тем самым устраняя проблемы с линейными моделями, которые бы мешали друг другу. Наконец, рассматриваются дополнительные факторы для разработки и производства персонализированных вакцин на основе неоантигенов.
КРАТКОЕ ОПИСАНИЕ НЕСКОЛЬКИХ ВИДОВ ГРАФИЧЕСКИХ МАТЕРИАЛОВ
[0010] Эти и другие характеристики, аспекты и преимущества данного изобретения станут более понятными в отношении нижеследующего описания и сопроводительных графических материалов, в которых:
[0011] На фигуре (фиг.) 1А изображены современные клинические подходы к идентификации неоантигенов.
[0012] На фиг. 1В изображено, что <5% спрогнозированных связанных пептидов презентированы на опухолевых клетках.
[0013] На фиг. 1С изображено влияние проблемы специфичности прогнозирования неоантигена.
[0014] На фиг. 1D изображено, что прогнозирование связывания недостаточно для идентификации неоантигенов.
[0015] На фиг. 1E изображена вероятность ГКГС-1 презентации в зависимости от длины пептида.
[0016] На фиг. 1F изображен пример пептидного спектра, генерируемого стандартом динамического диапазона Promega.
[0017] На фиг. 1G изображено, как добавление признаков увеличивает прогностическую ценность положительного результата модели.
[0018] На фиг. 2А представлен обзор среды для определения вероятностей презентации пептидов у пациентов в соответствии с вариантом осуществления.
[0019] На фиг. 2В и 2С изображен способ получения информации о презентации в соответствии с вариантом осуществления.
[0020] На фиг. 3 представлена высокоуровневая блок-схема, изображающая компоненты компьютерной логики системы идентификации презентации, в соответствии с одним вариантов осуществления.
[0021] На фиг. 4 изображен иллюстративный совокупность данных для обучения в соответствии с одним вариантом осуществления.
[0022] На фиг. 5 изображена иллюстративная сетевая модель в ассоциации с аллелем ГКГС.
[0023] На фиг. 6 изображена иллюстративная сетевая модель, совместно используемая аллелями ГКГС.
[0024] На фиг. 7 изображено генерирование вероятности презентации для пептида в ассоциации с аллелем ГКГС с использованием иллюстративной сетевой модели.
[0025] На фиг. 8 изображено генерирование вероятности презентации для пептида в ассоциации с аллелем ГКГС с использованием иллюстративных сетевых моделей.
[0026] На фиг. 9 изображено генерирование вероятности презентации для пептида в ассоциации с аллелями ГКГС с использованием иллюстративных сетевых моделей.
[0027] На фиг. 10 изображено генерирование вероятности презентации для пептида в ассоциации с аллелями ГКГС с использованием иллюстративных сетевых моделей.
[0028] На фиг. 11 изображено генерирование вероятности презентации для пептида в ассоциации с аллелями ГКГС с использованием иллюстративных сетевых моделей.
[0029] На фиг. 12 изображено генерирование вероятности презентации для пептида в ассоциации с аллелями ГКГС с использованием иллюстративных сетевых моделей.
[0030] На фиг. 13 изображены результаты тестирования эффективности различных иллюстративных моделей презентации.
[0031] На фиг. 14 изображен пример компьютера для имплементации объектов, изображенных на фиг. 1 и 3.
ПОДРОБНОЕ ОПИСАНИЕ СУЩНОСТИ ИЗОБРЕТЕНИЯ
I. Определения
[0032] В общем, термины, используемые в формуле изобретения и описании изобретения, предназначены для толкования как имеющие общепринятое значение, понятное специалисту в данной области техники. Конкретные термины определены ниже для обеспечения дополнительной ясности. В случае конфликта между общепринятым значением и представленными определениями должны использоваться представленные определения.
[0033] Используемый в данном документе термин «антиген» представляет собой вещество, которое индуцирует иммунный ответ.
[0034] Используемый в данном документе термин «неоантиген» представляет собой антиген, который имеет по меньшей мере одно изменение, которое отличает его от соответствующего исходного антигена дикого типа, например, путем мутации в опухолевой клетке или посттрансляционной модификации, специфической для опухолевой клетки. Неоантиген может содержать полипептидную последовательность или нуклеотидную последовательность. Мутация может содержать инсерционно-делеционную мутацию со сдвигом или без сдвига рамки или считывания, миссенс- или нонсенс-замену, изменение сайта сплайсинга, геномную перестройку или слияние генов, или любые изменения генома или экспрессии, приводящие к нео-ОРС. Мутации также могут содержать сплайс-вариант.Посттрансляционные модификации, специфические к опухолевой клетке, могут содержать аберрантное фосфорилирование. Постгрансляционные модификации, специфические к опухолевой клетке, могут содержать генерируемый протеасомой сплайсированный антиген. См. Liepe et al., A large fraction of HLA class I ligands are proteasome-generated spliced peptides; Science. 2016 Oct 21; 354(6310):354-358.
[0035] Используемый в данном документе термин «неоантиген опухоли» представляет собой неоантиген, присутствующий в опухолевой клетке или ткани субъекта, но не в соответствующей нормальной клетке или ткани субъекта.
[0036] Используемый в данном документе термин «вакцина на основе неоантигена» представляет собой вакцинную конструкцию на основе одного или более неоантигенов, например, множества неоантигенов.
[0037] Используемый в данном документе термин «кандидатный неоантиген» представляет собой мутацию или другую аберрацию, приводящую к появлению новой последовательности, которая может представлять собой неоантиген.
[0038] Используемый в данном документе термин «кодирующая область» представляет собой часть(и) гена, которая кодирует белок.
[0039] Используемый в данном документе термин «кодирующая мутация» представляет собой мутацию, возникающую в кодирующей области.
[0040] Используемый в данном документе термин «ОРС» означает открытую рамку считывания.
[0041] Используемый в данном документе термин «НЕО-ОРС» представляет собой опухолеспецифическую ОРС, возникающую в результате мутации или другой аберрации, такой как сплайсинг.
[0042] Используемый в данном документе термин «миссенс-мутация» представляет собой мутацию, вызывающую замену одной аминокислоты на другую.
[0043] Используемый в данном документе термин «нонсенс-мутация» представляет собой мутацию, вызывающую замену аминокислоты на стоп-кодон.
[0044] Используемый в данном документе термин «мутация со сдвигом рамки считывания» представляет собой мутацию, вызывающую изменение в рамке считывания белка.
[0045] Используемый в данном документе термин «инсерционно-делеционная мутация» представляет собой инсерцию или делецию одной или более нуклеиновых кислот.
[0046] Используемый в данном документе термин «идентичность» в контексте двух или более последовательностей нуклеиновой кислоты или полипептида относится к двум или более последовательностям или подпоследовательностям, которые имеют определенный процент нуклеотидов или аминокислотных остатков, которые являются такими же, если сравнивать и выравнивать для максимального соответствия, как измерено с использованием одного из алгоритмов сравнения последовательностей, описанных ниже (например, BLASTP и BLASTN или других алгоритмов, доступных специалистам) или путем визуального контроля. В зависимости от приложения процент «идентичности» может быть больше области сравниваемой последовательности, например, больше, чем функциональный домен, или, альтернативно, существовать по всей длине двух последовательностей, подлежащих сравнению.
[0047] Для сравнения последовательностей, как правило, одна последовательность выполняет функцию эталонной последовательности, с которой сравниваются исследуемые последовательности. При использовании алгоритма сравнения последовательностей исследуемые и эталонные последовательности вводятся в компьютер, при необходимости обозначаются координаты подпоследовательности, и задаются программные параметры алгоритма для работы с последовательностями. Затем алгоритм сравнения последовательностей вычисляет идентичность последовательности в процентах для исследуемой последовательности(ов) относительно эталонной последовательности на основе заданных программных параметров. Альтернативно, сходство или разнородность последовательностей может быть установлена комбинированным присутствием или отсутствием конкретных нуклеотидов или, для транслированных последовательностей, аминокислот в выбранных положениях последовательностей (например, мотивы последовательностей).
[0048] Оптимальное выравнивание последовательностей для сравнения может быть проведено, например, с помощью алгоритма поиска локальной гомологии по Smith & Waterman, Adv. Appl. Math. 2:482 (1981), с помощью алгоритма выравнивания областей гомологии по Needleman & Wunsch, J. Mol. Biol. 48:443 (1970), с помощью способа поиска сходства по Pearson & Lipman, Proc. Nat'l. Acad. Sci. USA 85:2444 (1988), компьютеризированные реализации этих алгоритмов (GAP, BESTFIT, FASTA и TFASTA в пакете программ Wisconsin Genetics, Genetics Computer Group, 575 Science Dr., Мадисон, штат Висконсин), или путем визуального осмотра (см., как правило, Ausubel et al., ниже).
[0049] Одним из примеров алгоритма, который подходит для определения процентной идентичности последовательности и сходства последовательностей, является алгоритм BLAST, который описан в Altschul et al., J. Mol. Biol. 215:403-410 (1990). Программное обеспечение для проведения анализов BLAST доступно для общественности через Национальный центр биотехнологической информации.
[0050] Используемый в данном документе термин «непрерывное или сквозное прочитывание» представляет собой мутацию, вызывающую удаление естественного стоп-кодона.
[0051] Используемый в данном документе термин «эпитоп» представляет собой специфическую часть антигена, обычно связанного антителом или Т-клеточным рецептором.
[0052] Используемый в данном документе термин «иммуногенный» означает способность вызывать иммунный ответ, например, через Т-клетки, В-клетки или через те, и другие.
[0053] Используемый в данном документе термин «аффинность связывания HLA» (HLA - человеческий лейкоцитарный антиген) «аффинность связывания ГКГС» означает аффинность связывания между конкретным антигеном и специфическим аллелем ГКГС.
[0054] Используемый в данном документе термин «приманка» представляет собой зонд на основе нуклеиновой кислоты, используемый для обогащения специфической последовательности ДНК или РНК из образца.
[0055] Используемый в данном документе термин «вариант» представляет собой различие между нуклеиновыми кислотами субъекта и эталонным геномом человека, используемым в качестве контроля.
[0056] Используемый в данном документе термин «определение вариантов» является алгоритмическим определением наличия варианта, как правило, путем секвенирования.
[0057] Используемый в данном документе термин «полиморфизм» представляет собой вариант зародышевой линии, то есть вариант, обнаруженный во всех клетках, несущих ДНК индивидуума.
[0058] Используемый в данном документе термин «соматический вариант» представляет собой вариант, возникающий в клетках не зародышевой линии индивидуума.
[0059] Используемый в данном документе термин «аллель» представляет собой версию гена или версию генетической последовательности или версию белка.
[0060] Используемый в данном документе термин «тип HLA» является комплементом к аллелям генов HLA.
[0061] Используемый в данном документе термин «нонсенс-опосредованное разрушение» или «NMD» представляет собой деградацию мРНК клеткой из-за преждевременного стоп-кодона.
[0062] Используемый в данном документе термин «стволовая мутация» представляет собой мутацию, возникающую на ранней стадии развития опухоли и присутствующую в значительной части клеток опухоли.
[0063] Используемый в данном документе термин «субклональная мутация» представляет собой мутацию, возникающую на поздней стадии развития опухоли и присутствующую только в подсовокупности клеток опухоли.
[0064] Используемый в данном документе термин «экзом» представляет собой подсовокупность генома, кодирующего белки. Экзом может представлять собой совокупность экзонов генома.
[0065] Используемый в данном документе термин «логистическая регрессия» является регрессионной моделью для данных в двоичной форме из статистики, в которой логит-преобразование вероятности, используемое как зависимая переменная равная единице, моделируется как линейная функция зависимых переменных.
[0066] Используемый в данном документе термин «нейронная сеть» представляет собой модель машинного обучения для классификации или регрессии, состоящую из нескольких слоев линейных преобразований, за которыми следуют поэлементные нелинейности, обычно обученные посредством стохастического градиентного спуска и обратного распространения ошибки.
[0067] Используемый в данном документе термин «протеом» представляет собой совокупность всех белков, экспрессированных и/или транслированных клеткой, группой клеток или индивидуумом.
[0068] Используемый в данном документе термин «пептидома» представляет собой совокупность всех ГКГС-1- или ГКГС-II-презентированных пептидов на поверхности клетки. Пептидома может относится к свойству клетки или совокупности клеток (например, опухолевая пептидома, означающая объединение пептидомов всех клеток, которые содержат опухоль).
[0069] Используемый в данном документе термин «ELISPOT» означает метод иммуноферментных пятен, который является общим методом для мониторинга иммунных реакций у людей и животных.
[0070] Используемый в данном документе термин «декстрамеры» представляет собой пептид-ГКГС-мультимеры на основе декстрана, используемые для окрашивания антигенспецифических Т-клеток в проточной цитометрии.
[0071] Используемый в данном документе термин «толерантность или иммунная толерантность» представляет собой состояние отсутствие иммунной реакции к одному или более антигенам, например, аутоантигенам.
[0072] Используемый в данном документе термин «центральная толерантность» представляет собой толерантность, влияющую на тимус, или путем удаления аутореактивных Т-клеточных клонов, или путем стимулирования аутореактивных Т-клеточных клонов для дифференциации в иммуносупрессивные регуляторные Т-клетки (Treg).
[0073] Используемый в данном документе термин «периферическая толерантность» представляет собой толерантность, влияющую на периферию, путем подавления или активации аутореактивных Т-клеток, которые сохраняют центральную толерантность или стимулируют эти Т-клетки дифференцироваться в Treg.
[0074] Термин «образец» может включать одну клетку или множество клеток, или фрагменты клеток, или аликвоту жидкости организма, взятую у субъекта, с помощью средств, включая венопункцию, экскрецию, эякуляцию, массаж, биопсию, отобранный шприцем аспират, пробы лаважа, соскоба, хирургического разреза или вмешательства или других средств, известных в данной области техники.
[0075] Термин «субъект» охватывает клетку, ткань или организм, человека или не относящегося к человеку, будь то in vivo, ex vivo или in vitro, самца или самку. Термин субъект включает млекопитающих, включая людей.
[0076] Термин «млекопитающее» охватывает как людей, так и не относящихся к человеку млекопитающих и включает, но не ограничивается ими, людей, приматов, отличных от человека, собак, кошек, мышей, коров, лошадей и свиней.
[0077] Термин «клинический фактор» относится к определению состояния субъекта, например, активности или тяжести заболевания. «Клинический фактор» охватывает все маркеры состояния здоровья субъекта, включая маркеры, не связанные с отбором образца, и/или другие характеристики субъекта, такие как, без ограничения, возраст и пол. Клиническим фактором может быть оценка, значение или совокупность значений, которые могут быть получены при оценке образца (или популяции образцов) у субъекта или субъекта в определенном состоянии. Клинический фактор также может быть спрогнозирован при помощи маркеров и/или других параметров, таких как имитаторы экспрессии генов. Клинические факторы могут включать тип опухоли, подтип опухоли и анамнез курения.
[0078] Аббревиатуры: ГКГС: главный комплекс гистосовместимости; HLA: человеческий лейкоцитарный антиген или генный локус человеческого ГКГС; NGS: секвенирование нового поколения; PPV: прогностическое значение положительного значения; TSNA: опухолеспецифический неоантиген; FFPE: зафиксированный в формалине и залитый парафином; NMD: нонсенс-опосредованное разрушение; НМРЛ: немелкоклеточный рак легких; ДК: дендритная клетка.
[0079] Используемые в данном документе и в прилагаемой формуле изобретения, формы единственного числа включают формы множественного числа, если из контекста явно не следует иное.
[0080] Любые термины, прямо не указанные в данном документе, понимаются как имеющие значения, обычно связанные с ними, как понимается в области техники изобретения. Определенные термины обсуждаются в данном документе для обеспечения дополнительных указаний специалисту-практику в описании композиций, устройств, способов и подобных аспектов изобретения и способа их создания или применения. Понятно, что одно и то же можно сказать более чем одним способом. Следовательно, альтернативные формулировки и синонимы могут использоваться для любого одного или более терминов, обсуждаемых в данном документе. Не следует придавать значения тому, объясняется или обсуждается этот термин. Предусмотрены некоторые синонимы или заменяемые методы, материалы и тому подобное. Составление одного или более синонимов или эквивалентов не исключает использования других синонимов или эквивалентов, если только это не указано явно. Использование примеров, включая примеры терминов, предназначено только для иллюстративных целей и не ограничивает объем и значение аспектов данного изобретения.
[0081] Все ссылки, выданные патенты и патентные заявки, приведенные в основной части описания изобретения, тем самым включены в качестве ссылки во всей их полноте для всех целей.
II. Способы идентификации неоантигенов
[0082] В данном документе раскрываются способы идентификации неоантигенов из опухоли субъекта, которые имеют вероятность быть презентированными на поверхности клеток опухоли и/или могут быть иммуногенными. В качестве примера один из таких способов может включать следующие этапы: получение по меньшей мере одного из данных нуклеотидного секвенирования из экзома, транскриптома или всего генома опухоли из опухолевой клетки субъекта, причем данные нуклеотидного секвенирования опухоли используются для получения данных, представляющих пептидные последовательности каждой из совокупности неоантигенов, и при этом пептидная последовательность каждого неоантигена содержит по меньшей мере одно изменение, которое делает его отличным от соответствующей последовательности исходного пептида дикого типа; ввода пептидной последовательности каждого неоантигена в одну или более моделей презентации для создания совокупности числовых вероятностей, что каждый из неоантигенов презентирован одним или более аллелями ГКГС на клеточной поверхности опухоли опухолевой клетки субъекта или клеток, присутствующих в опухоли, совокупность числовых вероятностей была идентифицирована по меньшей мере на основе полученных данных масс-спектрометрии; и выбор подсовокупности из совокупности неоантигенов на основе совокупности числовых вероятностей для создания совокупности выбранных неоантигенов.
[0083] Модель презентации может включать статистическую регрессию или модель машинного обучения (например, глубокое обучение), обученную по совокупности эталонных данных (также называемую тренировочной совокупностью данных), содержащую совокупность соответствующих меток, причем совокупность эталонных данных, полученная из каждого множества отдельных субъектов, где необязательно некоторые субъекты могут иметь опухоль, и при этом совокупность эталонных данных включает по меньшей мере одно из следующего: данные, представляющие нуклеотидные последовательности экзома из опухолевой ткани, данные, представляющие нуклеотидные последовательности экзома из нормальной ткани, данные, представляющие нуклеотидные последовательности транскриптомы из опухолевой ткани, данные, представляющие последовательности протеома из опухолевой ткани, и данные, представляющие последовательности пептидома ГКГС из опухолевой ткани, и данные, представляющие последовательности пептидома ГКГС из нормальной ткани. Эталонные данные могут дополнительно содержать данные масс-спектрометрии, данные секвенирования, данные секвенирования РНК и данные протеомики для одноаллельных клеточных линий, модифицированных для экспрессии предварительно определенного аллеля ГКГС, которые затем подвергаются воздействию синтетических белков, нормальных и опухолевых клеточных линий человека, и свежих и замороженных первичных образцов, и Т-клеточных анализов (например, ELISPOT). В некоторых аспектах совокупность эталонных данных содержит каждую форму эталонных данных.
[0084] Модель презентации может содержать совокупность признаков, полученных по меньшей мере частично, из совокупности эталонных данных, причем совокупность признаков содержит по меньшей мере один из зависимых от аллеля признаков и независимых от аллеля признаков. В некоторых аспектах включают каждый признак.
[0085] Презентация дендритными клетками для признаков наивных Т-клеток может включать по меньшей мере один из следующих: признак, описанный выше. Дозу и тип антигена в вакцине, (например, пептид, мРНК, вирус и т.д.): (1) путь, по которому дендритные клетки (ДК) поглощают тип антигена (например, эндоцитоз, микропиноцитоз); и/или (2) эффективность, с которой антиген поглощается ДК. Дозу и тип адъюванта в вакцине. Длину последовательности антигена вакцины. Число и места введения вакцины. Исходное функционирование иммунной системы пациента (например, измеренное по истории последних инфекций, анализ крови и т.д.). Для РНК-вакцин: (1) интенсивность обмена белкого продукта мРНК в дендритной клетке; (2) скорость трансляции мРНК после поглощения дендритными клетками, измеренную в экспериментах in vitro или in vivo; и/или (3) количество или циклы трансляции мРНК после поглощения дендритными клетками, как измерено in vivo или in vitro. Присутствующие в пептиде мотивы расщепления протеазы, необязательно придающие дополнительный вес протеазам, обычно экспрессируются в дендритных клетках (как измерено с помощью секвенирования РНК или масс-спектрометрии). Уровень экспрессии протеасомы и иммунопротеасомы в типичных активированных дендритных клетках (которые могут быть измерены с помощью секвенирования РНК, масс-спектрометрии, иммуногистохимии или других стандартных методов). Уровни экспрессии специфического аллеля ГКГС у рассматриваемого индивидуума (например, измеренного с помощью секвенирования РНК или масс-спектрометрии), необязательно измеряются конкретно в активированных дендритных клетках или других иммунных клетках. Вероятность презентации пептида конкретным аллелем ГКГС у других индивидуумов, которые экспрессируют конкретный аллель ГКГС, необязательно измеряется специфически в активированных дендритных клетках или других иммунных клетках. Вероятность презентации пептидов аллелями ГКГС в одном и том же семействе молекул (например, HLA-A, HLA-B, HLA-C, HLA-DQ, HLA-DR, HLA-DP) у других индивидуумов, необязательно измеряется конкретно в активированных дендритных клетках или других иммунных клетках.
[0086] Функции ускользания от иммунной толерантности могут включать по меньшей мере одну из следующего: непосредственное определение собственной пептидомы методом белковой масс-спектрометрии выполняется по одному или нескольким типам клеток. Оценку собственной пептидомы путем объединения всех k-мерных (например, 5-25) подпоследовательностей собственных белков. Оценку собственной пептидомы с использованием модели презентации, аналогичной описанной выше модели презентации, применяемую ко всем немутационным собственным белкам, необязательно учитывающей варианты зародышевой линии.
[0087] Ранжирование может быть выполнено с использованием множества неоантигенов, обеспеченных по меньшей мере одной моделью, основанной по меньшей мере частично на числовых вероятностях. После ранжирования выбор может быть выполнен для выбора подсовокупности ранжированных неоантигенов в соответствии с критериями выбора. После выбора подсовокупность ранжированных пептидов может быть предоставлена в виде результатов.
[0088] Ряд совокупностей выбранных неоантигенов может составлять 20.
[0089] Модель презентации может представлять собой зависимость между наличием пары определенного одного из аллелей ГКГС и определенной аминокислоты в определенном положении пептидной последовательности; и вероятность презентации на поверхности опухолевых клеток, в частности, одним определенным ГКГС аллелем указанной пары, таким как пептидная последовательность, содержащая конкретную аминокислоту в конкретном положении.
[0090] Способ, раскрытый в данном документе, также может включать применение одной или более моделей презентации к пептидной последовательности соответствующего неоантигена для генерирования оценки зависимости для каждого из одного или более аллелей ГКГС, указывающих, будет ли аллель ГКГС представлять соответствующий неоантиген на основании по меньшей мере положения аминокислот пептидной последовательности соответствующего неоантигена.
[0091] Способ, раскрытый в данном документе, также может включать преобразование оценок зависимостей для генерирования соответствующей вероятности на аллель для каждого аллеля ГКГС, указывающего вероятность того, что соответствующий аллель ГКГС будет презентировать соответствующий неоантиген; и комбинирование вероятностей аллелей для генерации числовой вероятности.
[0092] Этап преобразования оценок зависимости может моделировать презентацию пептидной последовательности соответствующего неоантигена как взаимоисключающего.
[0093] Способ, раскрытый в данном документе, также может включать преобразование комбинации оценок зависимостей для генерирования численного значения вероятности.
[0094] Этап преобразования комбинации оценок зависимостей может моделировать презентацию пептидной последовательности соответствующего неоантигена как интерферирующего между аллелями ГКГС.
[0095] Совокупность числовых значений вероятностей может быть дополнительно идентифицирована по меньшей мере аллель-невзаимодействующим признаком, и способ, раскрытый в данном документе, может также включать применение аллель-невзаимодействующей модели из одной или более моделей презентациик аллель-невзаимодействующим признакам для генерации оценки зависимости для аллель-невзаимодействующих признаков, указывающих, будет ли презентирована пептидная последовательность соответствующего неоантигена на основе аллель-невзаимодействующих признаков.
[0096] Способ, раскрытый в данном документе, также может включать комбинирование оценки зависимости для каждого аллеля ГКГС в одном или более аллелях ГКГС с оценкой зависимости для аллель-невзаимодействующего признака; трансформирование комбинированные оценки зависимостей для каждого аллеля ГКГС, чтобы генерировать соответствующую вероятность для каждого аллеля для аллеля ГКГС, указывающее на вероятность того, что соответствующий аллель ГКГС будет презентировать соответствующий неоантиген; и комбинирование вероятностей для каждого аллеля для генерации числового значения вероятности.
[0097] Способ, раскрытый в данном документе, также может включать преобразование комбинации оценок зависимостей для каждого из аллелей ГКГС и оценки зависимости для аллель-невзаимодействующих признаков для генерирования численного значения вероятности.
[0098] Совокупность числовых параметров для модели презентации может быть обучена на основе совокупности данных для обучения, включающего по меньшей мере совокупность пептидных последовательностей для обучения, идентифицированных как присутствующие во множестве образцов, и один или более аллелей ГКГС, ассоциированных с каждой пептидной последовательностью для обучения, причем пептидные последовательности для обучения идентифицируют с помощью масс-спектрометрии на выделенных пептидах, элюированных из аллелей ГКГС, полученных из множества образцов.
[0099] Образцы могут также содержать клеточные линии, модифицированные для экспрессии одного аллеля ГКГС класса I или класса II.
[00100] Образцы могут также содержать клеточные линии, модифицированные для экспрессии множества аллелей ГКГС класса I или класса II.
[00101] Образцы могут также содержать клеточные линии человека, взятые или полученные от множества пациентов.
[00102] Образцы могут также содержать свежие или замороженные образцы опухоли, полученные от множества пациентов.
[00103] Образцы могут также содержать свежие или замороженные образцы ткани, полученные от множества пациентов.
[00104] Образцы также могут содержать пептиды, идентифицированные с использованием Т-клеточных анализов.
[00105] Совокупность данных для обучения может дополнительно содержать данные, связанные с: количеством пептидов совокупности пептидов для обучения, присутствующих в образцах; длиной пептида совокупности пептидов для обучения в образцах.
[00106] Совокупность данных для обучения может быть сгенерирована путем сравнения совокупности пептидных последовательностей для обучения посредством сравнительного анализа первичной структуры с базой данных, содержащей совокупность известных белковых последовательностей, причем совокупность последовательностей белков для обучения больше и содержит пептидные последовательности для обучения.
[00107] Совокупность данных для обучения может быть сгенерирована на основе выполнения или выполненного нуклеотидного секвенирования на клеточной линии для получения по меньшей мере одного из данных секвенирования экзома, транскриптома или всего генома из клеточной линии, причем данные секвенирования включают по меньшей мере одну нуклеотидную последовательность, содержащую изменение.
[00108] Совокупность данных для обучения может быть сгенерирована на основе получения по меньшей мере из стандартных данных нуклеотидного секвенирования экзома, транскриптома и всего генома из образцов нормальной ткани.
[00109] Совокупность данных для обучения может дополнительно включать данные, связанные с последовательностями протеома, связанными с образцами.
[00110] Совокупность данных для обучения может дополнительно включать данные, связанные с последовательностями пептидома ГКГС, связанными с образцами.
[00111] Совокупность данных для обучения может дополнительно включать данные, связанные с измерениями аффинности связывания пептида-ГКГС по меньшей мере для одного из выделенных пептидов.
[00112] Совокупность данных для обучения может дополнительно включать данные, ассоциированные с измерениями стабильности связывания пептида-ГКГС по меньшей мере для одного из выделенных пептидов.
[00113] Совокупность данных для обучения может дополнительно включать данные, связанные с транскриптомами, ассоциированными с образцами.
[00114] Совокупность данных для обучения может дополнительно включать данные, связанные с геномами, ассоциированными с образцами.
[00115] Пептидные последовательности для обучения могут иметь длину в диапазоне k-меров, где к находится между 8-15, включительно для ГКГС класса I или 9-30 включительно для ГКГС класса II.
[00116] Способ, раскрытый в данном документе, также может включать кодирование пептидной последовательности с использованием схемы прямого кодирования.
[00117] Способ, раскрытый в данном документе, также может включать кодирование пептидных последовательностей для обучения с использованием схемы прямого кодирования справа налево.
[00118] Способ лечения субъекта, имеющего опухоль, включающий выполнение этапов по п. 1, и дополнительно включающий получение противоопухолевой вакцины, содержащей совокупность выбранных неоантигенов, и введение противоопухолевой вакцины субъекту.
[00119] Также в данном документе раскрыты способы получения противоопухолевой вакцины, включающие этапы: получения по меньшей мере одного из данных нуклеотидного секвенирования экзома, транскриптомы или всего генома опухоли опухолевой клетки субъекта, причем данные нуклеотидного секвенирования опухоли используются для получения данных, представляющих последовательности пептидов каждой из совокупности неоантигенов, и при этом пептидная последовательность каждого неоантигена содержит по меньшей мере одну мутацию, которая делает ее отличной от соответствующей исходной пептидной последовательности дикого типа; ввода пептидной последовательности каждого неоантигена в одну или более моделей презентации для создания совокупности числовых значений вероятностей, что каждый из неоантигенов презентирован одним или более аллелями ГКГС на поверхности опухолевых клеток клетки опухоли субъекта, при этом совокупность числовых значений вероятностей была идентифицирована по меньшей мере на основе полученных данных масс-спектрометрии; и выбора подсовокупности из совокупности неоантигенов на основе совокупности числовых значений вероятностей для создания совокупности выбранных неоантигенов; и продуцирования или изготовления противоопухолевой вакцины, содержащей совокупность выбранных неоантигенов.
[00120] Также в данном документе раскрыта противоопухолевая вакцина, содержащая совокупность выбранных неоантигенов, выбранных путем осуществления способа, включающего: получение по меньшей мере одного из данных нуклеотидного секвенирования опухоли экзома, транскриптомы или полного генома опухолевой клетки субъекта, причем данные нуклеотидного секвенирования опухоли используются для получения данных, представляющих пептидные последовательности каждого из совокупности неоантигенов, и при этом пептидная последовательность каждого неоантигена содержит по меньшей мере одну мутацию, которая делает ее отличной от соответствующей исходной пептидной последовательности дикого типа; ввод пептидной последовательности каждого неоантигена в одну или более моделей презентации для создания совокупности числовых значений вероятностей так, что каждый из неоантигенов презентирован одним или более аллелями ГКГС на поверхности опухолевых клеток опухолевой клетки субъекта, совокупность числовых значения вероятностей была идентифицирована по меньшей мере на основе полученных данных масс-спектрометрии; и выбор подсовокупности из совокупности неоантигенов на основе совокупности числовых значений вероятностей для создания совокупности выбранных неоантигенов; и продуцирование или изготовление противоопухолевой вакцины, содержащей совокупность выбранных неоантигенов.
[00121] Противоопухолевая вакцина может содержать одну или более нуклеотидных последовательностей, полипептидную последовательность, РНК, ДНК, клетку, плазмиду или вектор.
[00122] Противоопухолевая вакцина может содержать один или более неоантигенов, презентированных на поверхности опухолевых клеток.
[00123] Противоопухолевая вакцина может содержать один или более неоантигенов, которые являются иммуногенными у субъекта.
[00124] Противоопухолевая вакцина может не содержать один или более неоантигенов, которые индуцируют аутоиммунный ответ против нормальной ткани у субъекта.
[00125] Противоопухолевая вакцина может содержать адъювант.
[00126] Противоопухолевая вакцина может содержать наполнитель.
[00127] Способ, раскрытый в данном документе, может также включать выбор неоантигенов, которые имеют повышенную вероятность быть презентированными на поверхности опухолевых клеток относительно невыбранных неоантигенов на основе модели презентации.
[00128] Способ, раскрытый в данном документе, может также включать выбор неоантигенов, которые имеют повышенную вероятность быть способными индуцировать опухолеспецифический иммунный ответ у субъекта относительно невыбранных неоантигенов на основе модели презентации.
[00129] Способ, раскрытый в данном документе, также может включать выбор неоантигенов, которые имеют повышенную вероятность того, что они имеют вероятность быть презентированными наивными Т-клетками с помощью профессиональных антигенпрзентирующих клеток (АПК) относительно невыбранных неоантигенов на основе модели презентации, необязательно, причем АПК представляет собой дендритную клетку (ДК).
[00130] Способ, раскрытый в данном документе, может также включать выбор неоантигенов, которые имеют уменьшенную вероятность быть подвергнутыми ингибированию посредством центральной или периферической толерантности по отношению к невыбранным неоантигенам на основе модели презентации.
[00131] Способ, раскрытый в данном документе, может также включать выбор неоантигенов, которые имеют уменьшенную вероятность быть способными индуцировать аутоиммунный ответ на нормальную ткань у субъекта относительно невыбранных неоантигенов на основе модели презентации.
[00132] Данные секвенирования нуклеиновой последовательности экзома или транскриптома могут быть получены путем проведения секвенирования на опухолевой ткани.
[00133] Секвенирование может представлять собой секвенирование нового поколения (NGS) или любой подход массивного параллельного секвенирования.
[00134] Совокупность числовых значений вероятностей может быть дополнительно идентифицирована по меньшей мере с ГКГС-аллель-взаимодействующими признаками, включающими по меньшей мере одно из следующего: спрогнозированную аффинность, с которой связываются аллель ГКГС и кодируемый неоантигеном пептид; спрогнозированную стабильность комплекса кодируемого неоантигеном пептида -ГКГС; последовательность и длину кодируемого неоантигеном пептида; вероятность презентации кодируемых неоантигеном пептидов, с аналогичной последовательностью в клетках от других индивидуумов, экспрессирующих конкретный аллель ГКГС, как оценивается с помощью протеомного масс-спектрометрического анализа или другими способами; уровни экспрессии специфического аллеля ГКГС у субъекта, о котором идет речь (например, измеряются с помощью секвенирования РНК или масс-спектрометрии); независимую вероятность презентации кодируемой неоантигеном полной пептидной последовательности специфическим аллелем ГКГС у других отдельных субъектов, которые экспрессируют конкретный аллель ГКГС; независимую вероятность презентации кодируемой неоантигеном полной пептидной последовательности аллелями ГКГС в одном и том же семействе молекул (например, HLA-A, HLA-B, HLA-C, HLA-DQ, HLA-DR, HLA-DP) в других отдельных субъектах.
[00135] Совокупность числовых значений вероятностей дополнительно идентифицируется при помощи по меньшей мере ГКГС-аллель-невзаимодействующих признаков, включающих по меньшей мере одно из: С- и N-концевых последовательностей, фланкирующих кодируемый неоантигеном пептид в его исходной белковой последовательности; наличия мотивов расщепления протеазой в кодируемом неоантигеном пептиде, необязательно оцененном в соответствии с экспрессией соответствующих протеаз в опухолевых клетках (как измерено с помощью РНК-секвенирования или масс-спектрометрии); интенсивности обмена исходного белка, измеренной в соответствующем типе клеток; длины исходного белка, необязательно с учетом конкретных вариантов сплайсинга («изоформ»), наиболее высоко экспрессируемых в опухолевых клетках, как измерено с помощью РНК-секвенирования или масс-спектрометрии протеома, или как спрогнозировано из аннотации сплайсинговых мутаций соматических или зародышевой линии, обнаруженных в данных последовательностей ДНК или РНК; уровня экспрессии протеасомы, иммунопротеасомы, тимопротеасомы или других протеаз в опухолевых клетках (которые могут быть измерены с помощью РНК-секвенирования, масс-спектрометрии протеома или иммуногистохимии); экспрессии исходного гена кодируемого неоантигеном пептида (например, измеренного с помощью РНК-секвенирования или масс-спектрометрии); типичной тканеспецифической экспрессии исходного гена кодируемого неоантигеном пептида на разных стадиях клеточного цикла; полного каталога характеристик исходного белка и/или его доменов, как можно найти в, например, uniProt или PDB http://www.rcsb.org/pdb/home/home.do; характеристик, описывающих свойства домена исходного белка, содержащего пептид, например: вторичную или третичную структуру (например, альфа-спираль против бета-листа); альтернативного сплайсинга; вероятности презентации пептидов из исходного белка исследуемого кодируемого неоантигеном пептида у других отдельных субъектов; вероятности того, что пептид не будет обнаружен или сверхпрезентирован при помощи масс-спектрометрии из-за технических ошибок; экспрессии различных генных модулей/путей, измеренных при помощи РНК-секвенирования (которые не обязательно содержат исходный белок пептида), которые являются информативными относительно состояния опухолевых клеток, стромы или инфильтрирующих опухоли лимфоцитов (TIL); числа копий исходного гена кодируемого неоантигеном пептида в опухолевых клетках; вероятности того, что пептид связывается с ТАР, или с измеренной или спрогнозированной аффинностью связывания пептида с ТАР; уровня экспрессии ТАР в опухолевых клетках (которые могут быть измерены с помощью РНК-секвенирования, масс-спектрометрии протеома, иммуногистохимии); присутствия или отсутствия опухолевых мутаций, включая, но не ограничиваясь ими: драйверных мутаций в известных генах-драйверах рака, таких как EGFR, KRAS, ALK, RET, ROS1, TP53, CDKN2A, CDKN2B, NTRK1, NTRK2, NTRK3 и генах, кодирующих белки, вовлеченные в механизмы презентации антигенов (например, В2М, HLA-A, HLA-B, HLA-C, ТАР-1, ТАР-2, ТАРВР, CALR, CNX, ERP57, HLA-DM, HLA-DMA, HLA-DMB, HLA-DO, HLA-DOA, HLA-DOBHLA-DP, HLA-DPA1, HLA-DPB1, HLA-DQ, HLA-DQA1, HLA-DQA2, HLA-DQB1, HLA-DQB2, HLA-DR, HLA-DRA, HLA-DRB1, HLA-DRB3, HLA-DRB4, HLA-DRB5 или любом из генов, кодирующих компоненты протеасомы или иммунопротеасомы). Пептидов, презентация которых связана с компонентом механизма презентации антигена, которым является мутация с утратой функции в опухоли, уменьшили вероятность презентации; присутствие или отсутствие функциональных полиморфизмов зародышевой линии, включая, но не ограничиваясь ими: в генах, кодирующих белки, участвующие в механизмах презентации антигена (например, В2М, HLA-A, HLA-B, HLA-C, ТАР-1, ТАР-2, ТАРВР, CALR, CNX, ERP57, HLA-DM, HLA-DMA, HLA-DMB, HLA-DO, HLA-DOA, HLA-DOBHLA-DP, HLA-DPA1, HLA-DPB1, HLA-DQ, HLA -DQA1, HLA-DQA2, HLA-DQB1, HLA-DQB2, HLA-DR, HLA-DRA, HLA-DRB1, HLA-DRB3, HLA-DRB4, HLA-DRB5 или любом из генов, кодирующих компоненты протеасомы или иммунопротеасомы); типа опухоли (например, НМРЛ, меланома); клинического подтипа опухоли (например, плоскоклеточный рак легких в противовес неплоскоклеточного); анамнеза курения; типичной экспрессии исходного гена пептида в соответствующем типе опухоли или клиническом подтипе, необязательно стратифицированными при помощи драйверной мутации.
[00136] По меньшей мере мутация может представлять собой инсерционно-делеционную мутацию со сдвигом или без сдвига рамки считывания, миссенс- или нонсенс-замену, изменение сайта сплайсинга, геномную перестройку или слияние генов, или любые изменения генома или экспрессии, приводящие к нео-ОРС.
[00137] Опухолевая клетка может быть выбрана из группы, состоящей из: рака легкого, меланомы, рака молочной железы, рака яичников, рака предстательной железы, рака почки, рака желудка, рака толстой кишки, рака яичек, рака головы и шеи, рака поджелудочной железы, рака мозга, В-клеточной лимфомы, острого миелогенного лейкоза, хронического миелогенного лейкоза, хронического лимфолейкоза и Т-клеточного лимфоцитарного лейкоза, немелкоклеточного рака легкого и мелкоклеточного рака легкого.
[00138] Способ, раскрытый в данном документе, также может включать получение противоопухолевой вакцины, содержащей совокупность выбранных неоантигенов или ее подсовокупности, необязательно дополнительно включающий введение противоопухолевой вакцины субъекту.
[00139] По меньшей мере один из неоантигенов в совокупности выбранных неоантигенов, когда он находится в полипептидной форме, может включать по меньшей мере одно из: аффинности связывания с ГКГС с величиной ИК50 менее 1000 нМ для полипептидов ГКГС 1-ого класса длиной 8-15, 8, 9, 10, 11, 12, 13, 14 или 15 аминокислот, наличия мотивов последовательности внутри или рядом с полипептидом в исходной белковой последовательности, способствующей расщеплению протеасом, и наличие мотивов последовательности, способствующих ТАР транспорту.
[00140] Также раскрыты в данном документе способы получения модели для идентификации одного или более неоантигенов, которые, вероятно, будут презентированы на поверхности опухолевых клеток опухолевой клетки, включающие этапы: получения масс-спектрометрических данных, содержащих данные, связанные с множеством выделенных пептидов, элюированных из главного комплекса гистосовместимости (ГКГС), полученного из множества образцов; получение совокупности данных для обучения путем по меньшей мере идентификации совокупности пептидных последовательностей для обучения, присутствующих в образцах, и одного или более ГКГС, ассоциированных с каждой пептидной последовательностью для обучения; обучение совокупности числовых параметров модели презентации с использованием совокупности данных для обучения, содержащей пептидные последовательности для обучения, модели презентации, обеспечивающей множество числовых значений вероятностей так, что пептидные последовательности из опухолевой клетки презентированы одним или более аллелями ГКГС на поверхности опухолевых клеток.
[00141] Модель презентации может представлять зависимость между: наличием определенной аминокислоты в определенном положении пептидной последовательности; и вероятностью презентации одним из аллелей ГКГС опухолевой клетки пептидной последовательности, содержащей определенную аминокислоту в конкретном положении.
[00142] Образцы могут также содержать клеточные линии, модифицированные для экспрессии одного аллеля ГКГС класса I или класса II.
[00143] Образцы могут также содержать клеточные линии, модифицированные для экспрессии множества аллелей ГКГС класса I или класса II.
[00144] Образцы могут также содержать клеточные линии человека, взятые или полученные от множества пациентов.
[00145] Образцы могут также содержать свежие или замороженные образцы опухоли, полученные от множества пациентов.
[00146] Образцы также могут содержать пептиды, идентифицированные с использованием Т-клеточных анализов.
[00147] Совокупность данных для обучения может дополнительно содержать данные, связанные с: количеством пептидов совокупности пептидов для обучения, присутствующих в образцах; длиной пептида из совокупности пептидов для обучения в образцах.
[00148] Способ, раскрытый в данном документе, также может включать получение совокупности белковых последовательностей для обучения на основе пептидных последовательностей для обучения путем сравнения совокупности пептидных последовательностей для обучения путем сравнительного анализа первичной структуры с базой данных, содержащей совокупность известных белковых последовательностей, при этом совокупность белковых последовательностей для обучения длиннее и содержит пептидные последовательности для обучения.
[00149] Способ, раскрытый в данном документе, также может включать выполнение или проведение масс-спектрометрии на клеточной линии для получения по меньшей мере одного из данных нуклеотидного секвенирования экзома, транскриптома или всего генома из клеточной линии, данные нуклеотидного секвенирования, содержащие по меньшей мере одну последовательность белка, содержащую мутации.
[00150] Способ, раскрытый в данном документе, также может включать кодирование пептидных последовательностей для обучения с использованием схемы прямого кодирования.
[00151] Способ, раскрытый в данном документе, также может включать получение по меньшей мере одного из данных стандартного нуклеотидгного секвенирования экзома, транскриптома и всего генома из образцов нормальной ткани; и обучение совокупности параметров модели презентации с использованием данных стандартного нуклеотидного секвенирования.
[00152] Совокупность данных для обучения может дополнительно включать данные, связанные с последовательностями протеома, ассоциированными с образцами.
[00153] Совокупность данных для обучения может дополнительно включать данные, связанные с последовательностями пептидома ГКГС, ассоциированными с образцами.
[00154] Совокупность данных для обучения может дополнительно включать данные, ассоциированные с измерениями аффинности связывания пептида-ГКГС по меньшей мере для одного из выделенных пептидов.
[00155] Совокупность данных для обучения может дополнительно включать данные, ассоциированные с измерениями стабильности связывания пептида-ГКГС по меньшей мере для одного из выделенных пептидов.
[00156] Совокупность данных для обучения может дополнительно включать данные, связанные с транскриптомами, ассоциированными с образцами.
[00157] Совокупность данных для обучения может дополнительно включать данные, связанные с геномами, ассоциированными с образцами.
[00158] Способ, раскрытый в данном документе, может также включать логическое регрессирование совокупности параметров.
[00159] Пептидные последовательности для обучения могут иметь длину в диапазоне k-меров, где к находится между 8-15, включительно для ГКГС класса I или 9-30 включительно для ГКГС класса II.
[00160] Способ, раскрытый в данном документе, также может включать кодирование пептидных последовательностей для обучения с использованием схемы прямого кодирования справа налево.
[00161] Способ, раскрытый в данном документе, может также включать определение значений для совокупности параметров с использованием алгоритма глубокого обучения.
[00162] В данном документе раскрыты способы идентификации одного или более неоантигенов, которые имеют вероятность быть презентированными на поверхности опухолевых клеток опухолевой клетки, включающие выполнение этапов: получения масс-спектрометрических данных, содержащих данные, связанные с множеством выделенных пептидов, элюированных из главного комплекса гистосовместимости (ГКГС), полученных из множества свежих или замороженных образцов опухоли; получения совокупности данных для обучения по меньшей мере идентификации совокупности пептидных последовательностей для обучения, присутствующих в образцах опухоли, и презентированных на одном или более аллелях ГКГС, ассоциированных с каждой пептидной последовательностью для обучения; получения совокупности белковых последовательностей для обучения на основе пептидных последовательностей для обучения; и обучения совокупности числовых параметров модели презентации с использованием белковых последовательностей для обучения и пептидных последовательностей для обучения, модели презентации, обеспечивающей множество числовых значений вероятностей так, что пептидные последовательности из опухолевой клетки презентированы одним или более аллелями ГКГС на поверхности опухолевой клетки.
[00163] Модель презентации может представлять собой зависимость между наличием пары определенного одного из аллелей ГКГС и определенной аминокислоты в определенном положении пептидной последовательности; и вероятностью презентации на поверхности опухолевых клеток, в частности, одним определенным ГКГС аллелем указанной пары, такой пептидной последовательности, содержащей конкретную аминокислоту в конкретном положении.
[00164] Способ, раскрытый в данном документе, также может включать выбор подсовокупности неоантигенов, в котором подсовокупность неоантигенов выбирают, поскольку каждый из них имеет повышенную вероятность того, что он презентирован на клеточной поверхности опухоли относительно одного или более различных неоантигенов опухолей.
[00165] Способ, раскрытый в данном документе, также может включать выбор подсовокупности неоантигенов, в котором подсовокупность неоантигенов выбирают, поскольку каждый из них имеет повышенную вероятность того, что он способен индуцировать опухолеспецифический иммунный ответ у субъекта относительно одного или более различных неоантигенов опухолей.
[00166] Способ, раскрытый в данном документе, также может включать выбор подсовокупности неоантигенов, в котором подсовокупность неоантигенов выбирают, поскольку каждый из них имеет повышенную вероятность того, что он может быть презентирован наивным Т-клеткам с помощью профессиональных антигенпрезентирующих клеток (АПК) относительно одного или более различных неоантигенов опухоли, при этом, необязательно, АПК представляет собой дендритную клетку (ДК).
[00167] Способ, раскрытый в данном документе, также может включать выбор подсовокупности неоантигенов, в котором подмсовокупность неоантигенов выбирают потому, что каждый из них имеет уменьшенную вероятность того, что он подвержен ингибированию посредством центральной или периферической толерантности относительно одного или более различных неоантигенов опухолей.
[00168] Способ, раскрытый в данном документе, также может включать выбор подсовокупности неоантигенов, в котором подсовокупность неоантигенов выбирают, потому что каждый из них имеет уменьшенную вероятность того, что он способен индуцировать аутоиммунный ответ на нормальную ткань у субъекта относительно одного или более различных неоантигенов опухолей.
[00169] Способ, раскрытый в данном документе, также может включать выбор подсовокупности неоантигенов, в котором подсовокупность неоантигенов выбирают, поскольку каждый из них имеет уменьшенную вероятность того, что он будет избирательно посттрансляционно модифицирован в опухолевых клетках по сравнению с АПК, при этом, необязательно, АПК представляет собой дендритную клетку (ДК).
[00170] Применение способов в данном документе будет использовать, если не указано иное, обычные способы химии белков, биохимии, методов рекомбинантной ДНК и фармакологии, в соответствии с уровнем техники. Такие методы полностью описываются в литературе. См., например, Т.Е. Creighton, Proteins: Structures and Molecular Properties (W.H. Freeman and Company, 1993); A.L. Lehninger, Biochemistry (Worth Publishers, Inc., current addition); Sambrook, et al., Molecular Cloning: A Laboratory Manual (2nd Edition, 1989); Methods In Enzymology (S. Colowick and N. Kaplan eds., Academic Press, Inc.); Remington's Pharmaceutical Sciences, 18th Edition (Easton, Pennsylvania: Mack Publishing Company, 1990); Carey and Sundberg Advanced Organic Chemistry 3rd Ed. (Plenum Press) Vols A and B(1992).
III. Идентификация опухолеспецифических мутаций в неоантигенах
[00171] В данном документе также раскрыты способы идентификации определенных мутаций (например, варианты или аллели, которые присутствуют в раковых клетках). В частности, эти мутации могут присутствовать в геноме, транскриптоме, протеоме или экзоме раковых клеток у субъекта, имеющего рак, но не в нормальной ткани у субъекта.
[00172] Генетические мутации в опухолях можно считать полезными для иммунологического нацеливания опухолей, если они приводят к изменениям в аминокислотной последовательности белка исключительно в опухоли. Пригодные мутации включают: (1) несинонимические мутации, приводящие к различным аминокислотам в белке; (2) мутации с прочитанным терминатором, в которых стоп-кодон модифицирован или удален, что приводит к трансляции более длинного белка с новой опухолеспецифической последовательностью на С-конце; (3) мутации сайта сплайсинга, которые приводят к включению интрона в зрелую мРНК и, таким образом, уникальную опухолеспецифическую последовательность белка; (4) хромосомные перестройки, которые приводят к образованию химерного белка с опухолеспецифическими последовательностями на стыке 2 белков (например, слияние генов); (5) мутации или делеции со сдвигом рамки считывания, которые приводят к новой открытой рамке считывания с новой опухолеспецифической белковой последовательностью. Мутации может также содержать одну или более инсерционно-делеционные мутации без сдвига рамки считывания, миссенс- или нонсенс-замену, изменение сайта сплайсинга, геномную перестройку или слияние генов, или любые изменения генома или экспрессии, приводящие к нео-ОРС.
[00173] Пептиды с мутациями или мутантными полипептидами, возникающие, например, из сплайсинга, сдвига рамки считывания, или мутации слияния генов в опухолевых клетках, могут быть идентифицированы путем секвенирования ДНК, РНК или белка в опухоли в противоположность нормальных клеток.
[00174] Также мутации могут включать ранее идентифицированные опухолеспецифические мутации. Известные опухолевые мутации можно найти в базе данных каталога соматических мутаций в раке (COSMIC).
[00175] Для обнаружения наличия определенной мутации или аллеля в ДНК или РНК человека доступны различные способы. Прогресс в данной области обеспечил точный, легкий и недорогой крупномасштабное генотипирование SNP (однонуклеотидных полиморфизмов). Например, было описано несколько методов, включая динамическую аллелеспецифическую гибридизацию (DASH), микропланшетный диагональный электрофорез в геле (MADGE), пиросеквенирование, олигонуклеотид-специфическое лигирование, систему TaqMan, а также различные ДНК-чип-технологии, такие как SNP-чипы Affymetrix. Эти методы используют амплификацию целевой генетической области, как правило, с помощью ПЦР. Другие методы, основанные на генерации малых сигнальных молекул путем инвазивного расщепления с последующей масс-спектрометрией или иммобилизованными замыкающими кольцо зондами и амплификацией по типу катящегося кольца. Ниже излагаются некоторые из методов, известных в данной области техники для определения конкретных мутаций.
[00176] Способы обнаружения на основе ПЦР могут включать мультиплексную амплификацию множества маркеров одновременно. Например, в данной области техники хорошо известно, что ПЦР-праймеры для получения ПЦР-продуктов, которые не перекрываются по размеру, могут быть проанализированы одновременно. Альтернативно, можно амплифицировать различные маркеры с помощью праймеров, которые дифференциально помечены и, таким образом, каждый может быть дифференциально обнаружен. Конечно, способы обнаружения, основанные на гибридизации, обеспечивают дифференциальное обнаружение нескольких ПЦР-продуктов в образце. В данной области техники известны другие способы, обеспечивающие мультиплексный анализ множества маркеров.
[00177] Для облегчения анализа однонуклеотидных полиморфизмов в геномной ДНК или клеточной РНК были разработаны несколько методов. Например, однонуклеотидный полиморфизм может быть обнаружен с использованием специального резистентного к экзонуклеазе нуклеотида, как описано, например, в Mundy, С.R. (Патент США №4656127). Согласно способу, праймер, комплементарный аллельной последовательности непосредственно в 3'' к полиморфному сайту, можно гибридизировать с целевой молекулой, полученной от конкретного животного или человека. Если полиморфный сайт на целевой молекуле содержит нуклеотид, который является комплементарным конкретному присутствующему нуклеотидному производному, резистентному экзонуклеазе, то это производное будет включено в конец гибридизированного праймера. Такое включение делает праймер устойчивым к экзонуклеазе и тем самым обеспечивает его обнаружение. Так как идентичность резистентного к экзонуклеазе производного образца известна, обнаружение того, что праймер стал устойчивым к экзонуклеазам, демонстрирует, что нуклеотид(ы), присутствующий в полиморфном сайте целевой молекулы, комплементарен нуклеотидному производному, используемому в реакции. Преимущество этого метода состоит в том, что он не требует определения большого количества излишних данных последовательности.
[00178] Метод на основе растворов может быть использован для определения идентичности нуклеотида полиморфного сайта. Cohen, D. et al. (патент Франции 2650840; заявка РСТ № WO 91/02087). Как и в методе Mundy в патенте США №4656127 используется праймер, который комплементарен аллельным последовательностям непосредственно в 3'' к полиморфному сайту. Метод определяет идентичность нуклеотида этого сайта с использованием меченых производных дидезоксинуклеотида, которые, если комплементарны нуклеотиду полиморфного сайта, будут включены в конец праймера.
[00179] Альтернативный метод, известный как генетический битанализ или GBA, описан Goelet, P. et al. (заявка РСТ №92/15712). Метод Goelet, P. et al. использует смеси меченых терминаторов и праймер, который комплементарен последовательности 3'' в полиморфном сайте. Меченый терминатор, который включен, таким образом, определяется при помощи и комплементарен нуклеотиду, присутствующему в полиморфном сайте целевой молекулы, которую оценивают.В отличие от метода Cohen et al. (патент Франции 2650840; заявка РСТ № WO 91/02087), метод Goelet, P. et al. может быть анализом гетерогенных фаз, при котором праймер или целевую молекулу иммобилизуют на твердой фазе.
[00180] Было описано несколько процедур введения праймер-управляемых нуклеотидов для анализа полиморфных сайтов в ДНК (Komher, J.S. et al., Nucl. Acids. Res. 17:7779-7784 (1989); Sokolov, В.P., Nucl. Acids Res. 18:3671 (1990); Syvanen, A.-C, et al., Genomics 8:684-692 (1990); Kuppuswamy, M.N. et al., Proc. Natl. Acad. Sci. (U.S.A.) 88:1143-1147 (1991); Prezant, T.R. et al., Hum. Mutat. 1:159-164 (1992); Ugozzoli, L. et al., GATA 9:107-112 (1992); Nyren, P. et al., Anal. Biochem. 208:171-175 (1993)). Данные методы отличаются от GBA тем, что они используют включение меченых дезоксинуклеотидов для различения оснований в полиморфном сайте. В таком формате, поскольку сигнал пропорционален количеству введенных дезоксинуклеотидов, полиморфизмы, которые возникают в прогонах того же нуклеотида, могут приводить к сигналам, которые пропорциональны длительности прогона (Syvanen, A.-C, et al., Амер. J. Hum. Genet. 52:46-59 (1993)).
[00181] Ряд способов получает информацию о последовательности непосредственно от миллионов отдельных молекул ДНК или РНК параллельно. Технологии одномолекулярного секвенирования путем синтеза в реальном времени основаны на обнаружении флуоресцентных нуклеотидов, поскольку они включены в находящуюся в процессе образования цепь ДНК, которая комплементарна последовательности, которая секвенирована. В одном способе олигонуклеотиды длиной 30-50 оснований ковалентно заякорены на 5'-конце на покровные стекла. Эти заякоренные цепи выполняют две функции. Во-первых, они действуют как сайты захвата для целевых матричных цепей, если матрицы сконфигурированы с хвостами захвата, комплементарными олигонуклеотидам, связанным с поверхностью. Они также действуют как праймеры для матрицы, направленного удлинения праймера, который составляет основу для считывания последовательности. Праймеры захвата функционируют как сайт фиксированного положения для определения последовательности с использованием нескольких циклов синтеза, обнаружения и химического расщепления красителя-линкера для удаления красителя. Каждый цикл состоит из добавления полимеразы/смеси меченых нуклеотидов, промывки, визуализации и расщепления красителя. В альтернативном способе полимеразу модифицируют молекулой флуоресцентного донора и иммобилизуют на предметном стекле, тогда как каждый нуклеотид кодируется цветом с акцепторной флуоресцентной частью, присоединенной к гамма-фосфату. Система обнаруживает взаимодействие между флуоресцентно меченной полимеразой и флуоресцентно модифицированным нуклеотидом, поскольку нуклеотид становится включенным в цепь de novo. Существуют и другие технологии секвенирования путем синтеза.
[00182] Для идентификации мутаций можно использовать любую подходящую платформу для секвенирования путем синтеза. Как описано выше, в настоящее время доступны четыре основные платформы для секвенирования путем синтеза: геномные секвенаторы от Roche/454 Life Sciences, анализатор 1G от Illumina/Solexa, система SOLiD от Applied BioSystems и система Heliscope от Helicos Biosciences. Платформы для секвенирования путем синтеза также были описаны Pacific Biosciences и VisiGen Biotechnologies. В некоторых вариантах осуществления множество секвенированных молекул нуклеиновой кислоты связаны с носителем (например, твердой подложкой). Для иммобилизации нуклеиновой кислоты на носителе на 3'- и/или 5-конце матрицы может быть добавлена последовательность захвата/универсальный затравочный сайт. Нуклеиновые кислоты могут быть связаны с носителем гибридизацией последовательности захвата с комплементарной последовательностью, ковалентно связанной с носителем. Последовательность захвата (также называемая универсальной последовательностью захвата) представляет собой последовательность нуклеиновой кислоты, комплементарную последовательности, прикрепленной к носителю, который может в обычном порядке служить универсальным праймером.
[00183] В качестве альтернативы последовательности захвата член связывающей пары (такой как, например, антитело/антиген, рецептор/лиганд или пара авидин-биотин, как описано, например, в заявке на патент США №2006/0252077), может быть связанным с каждым фрагментом, который должен быть захвачен на поверхности, покрытой соответствующим вторым элементом этой связывающей пары.
[00184] После захвата последовательность может быть проанализирована, например, путем детектирования отдельных молекул/секвенирования с помощью одной молекулы, например, как описано в примерах и в патенте США №№7283337, включая зависимое от матриц секвенирование путем синтеза. При секвенировании путем синтеза поверхностно-связанная молекула подвергается воздействию множества меченых нуклеотидных трифосфатов в присутствии полимеразы. Последовательность матрицы определяется порядком меченых нуклеотидов, включенных в 3'-конец растущей цепи. Это можно сделать в реальном времени или выполнить в режиме последовательной шаговой мультипликации. Для анализа в реальном времени могут быть включены различные оптические метки для каждого нуклеотида, и для стимуляции включенных нуклеотидов можно использовать несколько лазеров.
[00185] Секвенирование также может включать другие методы и платформы для массивного параллельного секвенирования или секвенирования нового поколения (NGS). Дополнительными примерами методов и платформ для массивного параллельного секвенирования являются Illumina HiSeq или MiSeq, Thermo PGM или Proton, Рас Bio RS II или Sequel, Gene Gene Qiagen и Oxford Nanopore MinION. Могут использоваться дополнительные аналогичные технологии массивного параллельного секвенирования, а также будущие поколения этих технологий.
[00186] Любой тип клеток или ткань могут быть использованы для получения образцов нуклеиновой кислоты для использования в описанных в данном документе способах. Например, образец ДНК или РНК может быть получен из опухоли или жидкости организма, например, крови, полученной известными способами (например, венопункцией), или из слюны. Альтернативно, тесты на нуклеиновые кислоты можно проводить на сухих образцах (например, на волосах или на коже). Кроме того, образец может быть получен для секвенирования из опухоли, и другой образец может быть получен из нормальной ткани для секвенирования, при этом нормальная ткань относится к тому же типу ткани, что и опухоль. Образец может быть получен для секвенирования из опухоли, и другой образец может быть получен из нормальной ткани для секвенирования, при этом нормальная ткань относится к отличному от типа ткани опухоли.
[00187] Опухоли могут включать один или более рак легкого, меланому, рак молочной железы, рак яичников, рак предстательной железы, рак почек, рак желудка, рак толстой кишки, рак яичек, рак головы и шеи, рак поджелудочной железы, рак головного мозга, В-клеточную лимфому, острый миелолейкоз, хронический миелолейкоз, хронический лимфоцитарный лейкоз и Т-клеточный лимфоцитарный лейкоз, немелкоклеточный рак легкого и мелкоклеточный рак легкого.
[00188] Альтернативно, белковая масс-спектрометрия может быть использована для идентификации или подтверждения наличия мутированных пептидов, связанных с белками ГКГС, на опухолевых клетках. Пептиды могут быть элюированы кислотой из опухолевых клеток или из молекул HLA, которые иммунопреципитируются из опухоли, а затем идентифицируются с использованием масс-спектрометрии.
IV. Неоантигены
[00189] Неоантигены могут содержать нуклеотиды или полипептиды. Например, неоантиген может представлять собой последовательность РНК, которая кодирует полипептидную последовательность. Таким образом, неоантигены, полезные в вакцинах, могут содержать нуклеотидные последовательности или полипептидные последовательности.
[00190] В данном документе раскрыты выделенные пептиды, которые содержат опухолеспецифичные мутации, идентифицированные описанными в данном документе способами, пептиды, которые содержат известные опухолеспецифические мутации, и мутантные полипептиды или их фрагменты, идентифицированные способами, описанными в данном документе. Неоантигенные пептиды могут быть описаны в контексте их кодирующей последовательности, причем неоантиген содержит нуклеотидную последовательность (например, ДНК или РНК), которая кодирует соответствующую полипептидную последовательность.
[00191] Один или более полипептидов, кодируемых неоантигенной нуклеотидной последовательностью, могут включать по меньшей мере одно из следующего: аффинность связывания с ГКГС с величиной ИК50 менее 1000 нМ, для пептидов ГКГС 1-ого класса длиной 8-15, 8, 9, 10, 11, 12, 13, 14 или 15 аминокислот, присутствие мотивов последовательности внутри или вблизи пептида, способствующего расщеплению протеасом, и присутствие мотивов последовательности, способствующие ТАР транспорту.
[00192] Один или более неоантигенов имеют вероятность быть презентированными на поверхности опухоли.
[00193] Один или более неоантигенов могут быть иммуногенными у субъекта, имеющего опухоль, например, способны вызывать Т-клеточный ответ или В-клеточный ответ у субъекта.
[00194] Один или более неоантигенов, которые индуцируют аутоиммунный ответ у субъекта, могут быть исключены из рассмотрения в контексте генерации вакцины для субъекта, имеющего опухоль.
[00195] Размер по меньшей мере одной неоантигенной пептидной молекулы может составлять, но не ограничивается ими, около 5, около 6, около 7, около 8, около 9, около 10, около 11, около 12, около 13, около 14, около 15, около 16, около 17, около 18, около 19, около 20, около 21, около 22, около 23, около 24, около 25, около 26, около 27, около 28, около 29, около 30, около 31, около 32, около 33, около 34, около 35, около 36, около 37, около 38, около 39, около 40, около 41, около 42, около 43, около 44, около 45, около 46, около 47, около 48, около 49, около 50, около 60, около 70, около 80, около 90, около 100, около 110, около 120 или более аминокислотных остатков и любого диапазона, выводимого из вышеуказанного. В конкретных вариантах осуществления неоантигенные пептидные молекулы равны или составляют менее 50 аминокислот.
[00196] Неоантигенные пептиды и полипептиды могут представлять собой: для ГКГС класса I 15 остатков или менее по длине и обычно состоят из около от 8 до 11 остатков, в частности 9 или 10 остатков; для ГКГС класса II 15-24 остатков.
[00197] Если необходимо, более длинный пептид может быть сконструирован несколькими способами. В одном случае, когда вероятности презентации пептидов на аллелях HLA спрогнозированы или известны, более длинный пептид может состоять из любого: (1) отдельно презентированных пептидов с удлинениями в 2-5 аминокислот по отношению к N- и С-концу каждого соответствующего продукта гена; (2) конкатенации некоторых или всех презентированных пептидов с удлиненными последовательностями для каждого. В другом случае, когда секвенирование выявляет длинную (>10 остатков) неоэпитопную последовательность, присутствующую в опухоли (например, из-за сдвига рамки считывания, сквозного прочитывания или включения интрона, что приводит к новой последовательности пептида), более длинный пептид будет состоять из: (3) всего участка новых опухолеспецифических аминокислот - таким образом, минуя необходимость в селекции на основе вычислительного или in vitro тестирования самого сильного HLA-презентированного более короткого пептида. В обоих случаях использование более длинного пептида обеспечивают эндогенный процессинг клетками пациентов и может приводить к более эффективной презентации антигена и индукции Т-клеточных ответов.
[00198] Неоантигенные пептиды и полипептиды имеют вероятность быть презентированными на белке HLA. В некоторых аспектах неоантигенные пептиды и полипептиды презентированы на белке HLA с большей аффинностью, чем пептид дикого типа. В некоторых аспектах неоантигенный пептид или полипептид может иметь ИК50 по меньшей мере менее 5000 нМ, по меньшей мере менее 1000 нМ, по меньшей мере менее 500 нМ, по меньшей мере менее 250 нМ, по меньшей мере менее 200 нМ, по меньшей мере менее 150 нМ, по меньшей мере менее 100 нМ, по меньшей мере менее 50 нМ или менее.
[00199] В некоторых аспектах неоантигенные пептиды и полипептиды не индуцируют аутоиммунный ответ и/или вызывают иммунологическую толерантность при введении субъекту.
[00200] Также предложены композиции, содержащие по меньшей мере два или более неоантигенных пептида. В некоторых вариантах осуществления композиция содержит по меньшей мере два различных пептида. По меньшей мере два различных пептида могут быть получены из одного и того же полипептида. Под разными полипептидами подразумевается, что пептид изменяется по длине, аминокислотной последовательности или и тому и другому. Эти пептиды получены из любого полипептида, который, как известно, или как было обнаружено, содержит опухолеспецифическую мутацию. Подходящие полипептиды, из которых могут быть получены неоантигенные пептиды, могут быть найдены, например, в базе данных COSMIC. В COSMIC отбирается всесторонняя информация о соматических мутациях в раке человека. Пептид содержит опухолеспецифическую мутацию. В некоторых аспектах опухолеспецифическая мутация представляет собой драйверную мутацию для конкретного типа рака.
[00201] Неоантигенные пептиды и полипептиды, обладающие необходимой активностью или свойством, могут быть модифицированы с целью получения определенных необходимых признаков, например, улучшенных фармакологических характеристик, при одновременном увеличении или по меньшей мере сохранении практически всей биологической активности немодифицированного пептида для связывания необходимой молекулы ГКГС и активации соответствующей Т-клетки. Например, неоантигенный пептид и полипептиды могут подвергаться различным изменениям, таким как замены, или консервативные, или неконсервативные, причем такие изменения могут обеспечить определенные преимущества при их использовании, такие как улучшение связывания ГКГС, стабильность или презентация. Под консервативными заменами подразумевается замена аминокислотного остатка другим, который биологически и/или химически подобен, например, один гидрофобный остаток на другой, или один полярный остаток на другой. Замены включают комбинации, такие как Gly, Ala; Val, Ile, Leu, Met; Asp, Glu; Asn, Gln; Ser, Thr; Lys, Arg; и Phe, Tyr. Эффект отдельных аминокислотных замен также может быть исследован с использованием D-аминокислот.Такие модификации могут быть сделаны с использованием хорошо известных способов синтеза пептидов, как описано, например, Merrifield, Science 232:341-347 (1986), Barany & Merrifield, The Peptides, Gross & Meienhofer, eds. (N.Y., Academic Press), стр. 1-284 (1979); и Stewart & Young, Solid Phase Peptide Synthesis, (Rockford, Ill., Pierce), 2d Ed. (1984).
[00202] Модификации пептидов и полипептидов различными аминокислотными миметиками или не встречающимися в природе аминокислотами могут быть особенно полезными для повышения стабильности пептида и полипептида in vivo. Стабильность может быть проанализирована несколькими способами. Например, для проверки стабильности использовались пептидазы и различные биологические среды, такие как плазма и сыворотка человека. См., например, Verhoef et al., Eur. J. Drug Metab Pharmacokin. 11:291-302 (1986). Период полувыведения пептидов можно легко определить, используя анализ 25%-ной сыворотки человека (об./об.). Протокол, как правило, следующий. Объединенную человеческую сыворотку (тип АВ, инактивированную не нагреванием) делипидизируют путем центрифугирования перед использованием. Затем сыворотку разбавляют до 25% средой для культивирования тканей RPMI и используют для проверки стабильности пептидов. В заданные интервалы времени небольшое количество реакционного раствора удаляют и добавляют к 6%-ной водной трихлоруксусной кислоте или этанолу. Мутный реакционный образец охлаждают (4°С) в течение 15 минут, а затем центрифугируют для осаждения осажденных белков сыворотки. Присутствие пептидов затем определяют с помощью обращенно-фазовой ВЭЖХ, используя условия хроматографической стабильности.
[00203] Пептиды и полипептиды могут быть модифицированы для получения желаемых характеристик, отличных от улучшенного периода полувыведения в сыворотке. Например, способность пептидов индуцировать активность ЦТЛ (цитотоксический Т-лимфоцит) может быть усилена связыванием с последовательностью, которая содержит по меньшей мере один эпитоп, который способен индуцировать ответ Т-хелперной клетки. Иммуногенные пептиды/Т-хелперные конъюгаты могут быть связаны с помощью спейсерной молекулы. Спейсер обычно состоит из относительно небольших нейтральных молекул, таких как аминокислоты или миметики аминокислот, которые по существу незаряжены в физиологических условиях. Спейсеры обычно выбирают, например, из Ala, Gly или других нейтральных спейсеров неполярных аминокислот или нейтральных полярных аминокислот. Следует понимать, что необязательно присутствующий спейсер должен содержать одни и те же остатки и, следовательно, может быть гетеро- или гомо-олигомером. В случае присутствия, спейсер, как правило, будет представлен по меньшей мере одним или двумя остатками, более вероятно тремя- шестью остатками. Альтернативно, пептид может быть связан с Т-хелперным пептидом без спейсера.
[00204] Неоантигенный пептид может быть связан с Т-хелперным пептидом или непосредственно, или через спейсер или при аминогруппе, или на карбоксильном конце пептида. N-конец или неоантигенного пептида, или Т-хелперного пептида может быть ацилирован. Иллюстративные Т-хелперные пептиды включают столбнячный анатоксин 830-843, грипп 307-319, спорозоит малярии 382-398 и 378-389.
[00205] Белки или пептиды могут быть получены любым способом, известным специалистам в данной области техники, включая экспрессию белков, полипептидов или пептидов посредством стандартных молекулярно-биологических методов, выделение белков или пептидов из природных источников или химический синтез белков или пептидов. Последовательности нуклеотидов и белков, полипептидов и пептидов, соответствующие различным генам, были ранее раскрыты и могут быть найдены в компьютеризированных базах данных, известных специалистам в данной области техники. Одной из таких баз данных являются базы данных Genbank и GenPept Национального центра биотехнологической информации, расположенные на веб-сайте Национального института здоровья. Кодирующие области для известных генов могут быть амплифицированы и/или экспрессированы с использованием способов, описанных в данном документе, или как известно специалистам в данной области техники. Альтернативно, различные коммерческие препараты из белков, полипептидов и пептидов известны специалистам в данной области техники.
[00206] В дополнительном аспекте неоантиген содержит нуклеиновую кислоту (например, полинуклеотид), которая кодирует неоантигенный пептид или его часть. Полинуклеотид может представлять собой, например, ДНК, кДНК, ПНК, ЦНК (циркулирующая нуклеиновая кислота), РНК (например, мРНК), или одно- и/или двухцепочечные, или нативные или стабилизированные формы полинуклеотидов, такие как, например, полинуклеотиды с тиофосфатным остовом, или их комбинации, и он может содержать или не содержать интроны. Другой дополнительный аспект обеспечивает вектор экспрессии, способный экспрессировать полипептид или его часть. Векторы экспрессии для разных типов клеток хорошо известны в данной области техники и могут быть выбраны без излишнего экспериментирования. Как правило, ДНК вставляют в вектор экспрессии, такой как плазмида, в правильной ориентации и подходящую рамку считывания для экспрессии. При необходимости ДНК может быть связана с соответствующими нуклеотидными последовательностями контроля транскрипционной и трансляционной регуляции, распознаваемыми желаемым хозяином, хотя такие контроли обычно доступны в векторе экспрессии. Затем вектор вводится в хозяина посредством стандартных методов. Руководство можно найти, например, в Sambrook et al. (1989) Molecular Cloning, A Laboratory Manual, Cold Spring Harbor Laboratory, Cold Spring Harbor, N.Y.
IV. Композиции вакцин
[00207] Также в данном документе раскрывается иммуногенная композиция, например, композиция вакцин, способная повышать специфический иммунный ответ, например, опухолеспецифичный иммунный ответ.Композиции вакцин обычно содержат множество неоантигенов, например, выбранных с использованием способа, описанного в данном документе. Композиции вакцин можно также назвать вакцинами.
[00208] Вакцина может содержать от 1 до 30 пептидов, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 различных пептидов, 6, 7, 8, 9, 10 11, 12, 13 или 14 различных пептидов или 12, 13 или 14 различных пептидов. Пептиды могут содержать посттрансляционные модификации. Вакцина может содержать от 1 до 100 или более нуклеотидных последовательностей, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64, 65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99, 100 или более различных нуклеотидных последовательностей, 6, 7, 8, 9, 10 11, 12, 13 или 14 различных нуклеотидных последовательностей или 12, 13 или 14 различных нуклеотидных последовательностей. Вакцина может содержать от 1 до 30 неоантигенных последовательностей, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64, 65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99, 100 или более различных неоантигенных последовательностей, 6, 7, 8, 9, 10 11, 12, 13 или 14 различных неоантигенных последовательностей или 12, 13 или 14 различных неоантигенных последовательностей.
[00209] В одном варианте осуществления различные пептиды и/или полипептиды или нуклеотидные последовательности, кодирующие их, выбраны так, что пептиды и/или полипептиды способны связываться с различными молекулами ГКГС, такими как молекула ГКГС класса I. В некоторых аспектах одна композиция вакцины содержит кодирующую последовательность для пептидов и/или полипептидов, способных связываться с наиболее часто встречающимися молекулами ГКГС класса I. Следовательно, композиции вакцин могут содержать различные фрагменты, способные связывать с по меньшей мере 2 предпочтительными, по меньшей мере 3 предпочтительными или по меньшей мере 4 предпочтительными молекулами ГКГС класса I.
[00210] Композиция вакцины может быть способна повышать специфический ответ цитотоксических Т-клеток и/или специфический ответ хелперных Т-клеток.
[00211] Композиция вакцины может дополнительно содержать адъювант и/или носитель. Примеры полезных адъювантов и носителей приведены ниже. Композиция может быть связана с носителем, таким как, например, белок или антигенпрезентирующая клетка, такая как, например, дендритная клетка (ДК), способная презентировать пептид Т-клетке.
[00212] Адъюванты представляют собой любое вещество, примесь которого в композиции вакцины увеличивает или иным образом модифицирует иммунный ответ на неоантиген. Носители могут представлять собой каркасные структуры, например, полипептид или полисахарид, к которым может быть присоединен неоантиген. При необходимости адъюванты конъюгируются ковалентно или нековалентно.
[00213] Способность адъюванта повышать иммунный ответ на антиген обычно проявляется значительным или существенным повышением иммунноопосредованной реакции или уменьшением симптомов заболевания. Например, усиление гуморального иммунитета обычно проявляется в значительном увеличении титра антител, выращенных до антигена, и увеличение активности Т-клеток обычно проявляется в увеличении клеточной пролиферации или клеточной цитотоксичности или секреции цитокинов. Адъювант может также изменять иммунный ответ, например, путем изменения преимущественно гуморального или Т-хелперного ответа на преимущественно клеточный или Т-хелперный ответ.
[00214] Подходящие адъюванты включают, но не ограничиваются ими, 1018 ISS, алюмокалиевые квасцы, соли алюминия, Amplivax, AS15, BCG, СР-870,893, CpG7909, СуаА, dSLIM, GM-CSF, IC30, IC31, имиквимод, ImuFact IMP321, IS Patch, ISS, ISCOMATRIX, JuvImmune, LipoVac, MF59, монофосфориллипид А, монтанид IMS 1312, монтанид ISA 206, монтанид ISA 50V, монтанид ISA-51, OK-432, ОМ-174, OM-197-MP-EC, ONTAK, векторную систему PepTel, микрочастицы ПЛГ (полилактид-ко-гликолид), резиквимод, SRL172, вирусомы и другие вирусоподобные частицы, YF-17D, ловушку ФРЭС, R848, бета-глюкан, Pam3Cys, стимулятор QS21 Aquila (Aquila Biotech, Воркестер, Массачусетс, США), который получен из сапонина, микобактериальных экстрактов и синтетических бактериальных клеточных стенок, а также других запатентованных адъювантов, таких как Detox Ribi. Quil или Superfos. Адъюванты, такие как неполный адъювант Фрейнда или ГМ-КСФ, являются пригодными. Несколько иммунологических адъювантов (например, MF59), специфических для дендритных клеток, и их получение были описаны ранее (Dupuis М, et al., Cell Immunol. 1998; 186(1): 18-27; Allison A C; Dev Biol Stand. 1998; 92:3-11). Также могут использоваться цитокины. Некоторые цитокины было непосредственно связаны с воздействием миграции дендритных клеток на лимфоидные ткани (например, ФНО-альфа), ускоряющие созревание дендритных клеток в эффективные антигенпрезентирующие клетки для Т-лимфоцитов (например, ГМ-КСФ, ИЛ-1 и ИЛ-4) (патент США №5849589, конкретно включенный в данное описание посредством ссылки во всей его полноте) и действующие как иммуноадъюванты (например, ИЛ-12) (Gabrilovich D I, et al., J Immunother Emphasis Tumor Immunol. 1996 (6):414-418).
[00215] Сообщалось также, что иммуностимулирующие CpG-олигонуклеотиды усиливают действие адъювантов при вакцинации. Могут также использоваться другие TLR-связывающие молекулы (TLR-Toll-подобный рецептор), такие как РНК-связывающие TLR 7, TLR 8 и/или TLR 9.
[00216] Другие примеры пригодных адъювантов включают, но не ограничиваются ими, химически модифицированные CpG (например, CpR, Idera), поли(I: С) (например, поли: CI2U), не-CpG бактериальную ДНК или РНК, а также иммуноактивные малые молекулы и антитела, такие как циклофосфамид, сунитиниб, бевацизумаб, целебрекс, NCX-4016, силденафил, тадалафил, варденафил, сорафиниб, XL-999, СР-547632, пазопаниб, ZD2171, AZD2171, ипилимумаб, тремелимумаб и SC58175, которые могут действовать терапевтически и/или в качестве адъюванта. Количество и концентрация адъювантов и добавок могут быть легко определены квалифицированным специалистом без излишнего экспериментирования. Дополнительные адъюванты включают колониестимулирующие факторы, такие как гранулоцитарно-моноцитарный колониестимулирующий фактор (ГМ-КСФ, сарграмостим).
[00217] Композиция вакцины может содержать более одного отличного адъюванта. Кроме того, терапевтическая композиция может содержать любое адъювантное вещество, включающее любое из вышеуказанных или их комбинации. Также предполагается, что вакцину и адъювант можно вводить вместе или отдельно в любой подходящей последовательности.
[00218] Носитель (или наполнитель) может присутствовать независимо от адъюванта. Функция носителя может, например, состоять в увеличении молекулярной массы конкретного мутанта для повышения активности или иммуногенности, обеспечения стабильности, увеличения биологической активности или увеличения периода полувыведения в сыворотке. Кроме того, носитель может способствовать презентации пептидов Т-клеткам. Носитель может представлять собой любой подходящий носитель, известный специалисту в данной области техники, например, белок или антигенпрезентирующая клетка. Белок-носитель может представлять собой, но не ограничен, гемоцианин моллюска Megathura crenulata, белки сыворотки, такие как трансферрин, бычий сывороточный альбумин, человеческий сывороточный альбумин, тиреоглобулин или овальбумин, иммуноглобулины или гормоны, такие как инсулин или пальмитиновая кислота. Для иммунизации человека носитель обычно является физиологически приемлемым носителем, приемлемым для людей и безопасным. Однако столбнячный анатоксин и/или дифтерийный анатоксин являются подходящими носителями. Альтернативно, носитель может представлять собой декстраны, например, сефарозу.
[00219] Цитотоксические Т-клетки (ЦТЛ) распознают антиген в виде пептида, связанного с молекулой ГКГС, а не с самим интактным чужеродным антигеном. Сама молекула ГКГС находится на клеточной поверхности клетки, содержащей антиген. Таким образом, активация ЦТЛ возможна, если присутствует тримерный комплекс пептидного антигена, молекулы ГКГС и АПК. Соответственно, он может усилить иммунный ответ, если для активации ЦТЛ используют не только пептид, но если дополнительно добавляются АПК с соответствующей молекулой ГКГС. Поэтому в некоторых вариантах осуществления композиция вакцины дополнительно содержит по меньшей мере одну антигенпрезентирующую клетку.
[00220] Неоантигены также могут быть включены в вакцинные платформы на основе вирусных векторных вакцин, таких как вирус осповакцины, вирус оспы кур, самовоспроизводящиеся альфавирусы, марабавирусы, аденовирусы (см., например, Tatsis et al., Adenoviruses, Molecular Therapy (2004) 10, 616-629), или лентивируса, включая, но не ограничиваясь этим, второй, третий или гибридный лентивирус второго и третьего поколений и рекомбинантный лентивирус любого поколения, предназначенный для нацеливания на конкретные типы клеток или рецепторы (см., например, Hu et al., Immunization Delivered by Lentiviral Vectors for Cancer and Infectious Diseases, Immunol Rev. (2011) 239(1): 45-61, Sakuma et al., Lentiviral vectors: basic to translational, Biochem J. (2012) 443(3):603-18, Cooper et al., Rescue of splicing-mediated intron loss maximizes expression in lentiviral vectors containing the human ubiquitin С promoter, Nucl. Acids Res. (2015) 43 (1): 682-690, Zufferey et al., Self-Inactivating Lentivirus Vector for Safe and Efficient In Vivo Gene Delivery, J. Virol. (1998) 72 (12): 9873-9880). В зависимости от упаковывающей способности вышеупомянутых вакцинных платформ на основе вирусных векторов данный подход может доставлять одну или более нуклеотидных последовательностей, которые кодируют один или более неоантигенных пептидов. Последовательности могут быть фланкированы немутированными последовательностями, могут быть разделены линкерами или им может предшествовать одна или более последовательностей, нацеленных на субклеточный компартмент (см., например, Gros et al., Prospective identification of neoantigen-specific lymphocytes in the peripheral blood of melanoma patients, Nat Med. (2016) 22 (4):433-8, Stronen et al., Targeting of cancer neoantigens with donor-derived T cell receptor repertoires, Science. (2016) 352 (6291):1337-41, Lu et al., Efficient identification of mutated cancer antigens recognized by T cells associated with durable tumor regressions, Clin Cancer Res. (2014) 20(13):3401-10). При введении в организм хозяина инфицированные клетки экспрессируют неоантигены и тем самым вызывают иммунную реакцию хозяина (например, ЦТЛ) против пептида(ов). Векторы на основе вируса осповакцины и способы, пригодные для протоколов иммунизации, описаны, например, в патенте США №4722848. Другим вектором является BCG (бацилла Кальмета-Герена). Векторы на основе BCG описаны в Stover et al. (Nature 351:456-460 (1991)). Широкий спектр других векторов вакцины, пригодные для терапевтического введения или иммунизации неоантигенов, например, векторов на основе Salmonella typhi, и тому подобное, будут очевидны специалистам в данной области техники из приведенного в данном документе описания.
IV.A. Дополнительные обсуждения по разработке и производству вакцин
IV.A.1. Определение совокупности пептидов, охватывающих все опухолевые субклоны
[00221] Под стволовыми пептидами подразумевают те, которые презентированы всеми или большинством опухолевых субклонов, будут приоритетными для включения в вакцину.53 Необязательно, если нет стволовых пептидов, спрогнозированных для презентации и иммуногенных с высокой вероятностью, или если спрогнозировано количество стволовых пептидов, определяемых как презентированные и иммуногенные с большой вероятностью, достаточно мало, чтобы в вакцину можно было включить дополнительные нестволовые пептиды, тогда дополнительные пептиды могут быть приоритетными путем оценки количества и идентичности опухолевых субклонов и выбора пептидов, чтобы максимизировать количество опухолевых субклонов охватываемых вакциной.54
IV.A.2. Определение приоритета неоантигена
[00222] После того, как все вышеупомянутые фильтры для кандидатных неоантигенов применены, для включения в вакцину все еще могут быть доступны дополнительные неоантигены по сравнению с вакцинной технологией, которая обеспечивается. Кроме того, может сохраняться неопределенность в отношении различных аспектов анализа неоантигена, и могут существовать компромиссы между различными свойствами кандидатных вакцинных неоантигенов. Таким образом, вместо предопределенных фильтров на каждом этапе процесса выбора можно рассмотреть интегрированную многомерную модель, которая помещает кандидатные неоантигены в пространство с по меньшей мере следующими осями и оптимизирует выбор с использованием интегративного подхода.
1. Риск аутоиммунных реакций или толерантности (риск зародышевой линии) (обычно предпочтительнее низкий риск аутоиммунных реакций)
2. Вероятность артефакта секвенирования (обычно предпочтительнее более низкая вероятность артефакта)
3. Вероятность иммуногенности (обычно предпочтительнее более высокая вероятность иммуногенности)
4. Вероятность презентации (обычно предпочтительнее более высокая вероятность презентации)
5. Экспрессия гена (обычно предпочтительнее более высокий уровень экспрессии)
6. Охват генов HLA (большее количество молекул HLA, участвующих в презентации совокупности неоантигенов, может снизить вероятность того, что опухоль избежит иммунной атаки посредством снижения экспрессии или мутации молекул HLA)
V. Терапевтические и производственные способы
[00223] Также предлагается способ индуцирования опухолеспецифического иммунного ответа в субъекта, вакцинация против опухоли, лечение и/или облегчение симптома рака у субъекта путем введения субъекту одного или более неоантигенов, таких как множество неоантигенов, идентифицированных с использованием способов, описанных в данном документе.
[00224] В некоторых аспектах у субъекта был диагностирован рак или он подвержен риску развития рака. Субъект может представлять собой человека, собаку, кошку, лошадь или любое животное, которому необходим опухолеспецифический иммунный ответ. Опухоль может представлять собой любую солидную опухоль, например, молочной железы, яичника, простаты, легкого, почки, желудка, толстой кишки, яичка, головы и шеи, поджелудочной железы, головного мозга, меланомы и другие опухоли тканей органов и гематологические опухоли, такие как лимфомы и лейкозы, включая острый миелолейкозный лейкоз, хронический миелолейкоз, хронический лимфоцитарный лейкоз, Т-клеточный лимфоцитарный лейкоз и В-клеточные лимфомы.
[00225] Неоантиген можно вводить в количестве, достаточном для индуцирования ЦТЛ ответа.
[00226] Неоантиген можно вводить отдельно или в комбинации с другими терапевтическими агентами. Терапевтический агент представляют собой, например, химиотерапевтический агент, радиацию или иммунотерапию. Любое подходящее терапевтическое лечение для конкретного рака может быть введено.
[00227] Кроме того, субъекту может дополнительно вводиться антииммуносупрессивный/иммуностимулирующий агент, такой как ингибитор контрольной точки. Например, субъекту можно дополнительно вводить анти-CTLA антитело или анти-PD-1, или анти-PD-L1. Блокада CTLA-4 или PD-L1 антителами может усилить иммунный ответ на раковые клетки у пациента. В частности, показано, что блокада CTLA-4 эффективна при соблюдении схемы вакцинации.
[00228] Можно определить оптимальное количество каждого неоантигена, которое должно быть включено в композици вакцины, и оптимальную схему применения. Например, неоантиген или его вариант могут быть получены для внутривенной (в/в) инъекции, подкожной (п/к) инъекции, внутрикожной (в/к) инъекции, интраперитонеальной (и/п) инъекции, внутримышечной (в/м) инъекции (i). Способы инъекции включают п/к., в/к, и/п, в/м и в/в. Способы инъекции ДНК или РНК включают, например, в/к, п/к, и/п и в/в. Другие способы введения композиции вакцины известны специалистам в данной области техники.
[00229] Вакцина может быть составлена таким образом, чтобы выбор, число и/или количество неоантигенов, присутствующих в композиции, являются(ется) специфическими к ткани, раку и/или пациенту. Например, точный выбор пептидов может быть основан на паттернах экспрессии исходных белков в данной ткани. Выбор может зависеть от конкретного типа рака, стадии заболевания, более ранних схем лечения, иммунного статуса пациента и, конечно же, HLA-гаплотипа пациента. Кроме того, вакцина может содержать персонализированные компоненты в соответствии с личными потребностями конкретного пациента. Примеры включают изменение выбора неоантигенов в соответствии с экспрессией неоантигена у конкретного пациента или корректировки для повторных курсов лечения после первого цикла или схемы лечения.
[00230] Для композиции, которая будет использоваться в качестве вакцины для рака, неоантигены с аналогичными нормальными аутопептидами, которые экспрессируются в больших количествах в нормальных тканях, можно исключить из или они могут присутствовать в небольших количествах в композиции, описанной в данном документе. С другой стороны, если известно, что опухоль пациента экспрессирует большие количества определенного неоантигена, соответствующая фармацевтическая композиция для лечения этого рака может быть предоставлена в больших количествах и/или может быть включен более, чем один неоантиген, специфический для конкретного неоантигена, особенно, или пути данного неоантигена.
[00231] Композиции, содержащие неоантиген, можно вводить индивидууму, уже страдающему от рака. В терапевтических применениях композиции вводят пациенту в количестве, достаточном для того, чтобы вызвать эффективный ЦТЛ-ответ на опухолевый антиген и вылечить или хотя бы частично устранить симптомы и/или осложнения. Количество, достаточное для достижения этого, определяется как «терапевтически эффективная доза». Количества, эффективные для этого применения, будут зависеть, например, от состава, способа введения, стадии и тяжести заболевания, которое необходимо лечить, от массы и общего состояния здоровья пациента и от решения лечащего врача. Следует иметь в виду, что композиции обычно можно использовать при серьезных болезненных состояниях, то есть в опасных для жизни или потенциально опасных для жизни ситуациях, особенно при метастазировании рака. В таких случаях, учитывая минимизацию примесей и относительную нетоксичную природу неоантигена, возможно и может быть желательно, чтобы лечащий врач вводил значительно превышающие нормы этих композиций.
[00232] Для терапевтического применения введение может начаться при обнаружении или хирургическом удалении опухолей. Затем следует повышение дозы до тех пор, пока по меньшей мере симптомы значительно ослабятся и в течение периода после этого.
[00233] Фармацевтические композиции (например, композиции вакцин) для терапевтического лечения предназначены для парентерального, местного, назального, перорального или локального применения. Фармацевтические композиции можно вводить парентерально, например, внутривенно, подкожно, внутрикожно или внутримышечно. Композиции можно вводить в месте хирургическое вмешательства, чтобы индуцировать местный иммунный ответ на опухоль. В данном документе раскрыты композиции для парентерального введения, которые содержат раствор неоантигенной и композиции вакцины, растворяют или суспендируют в приемлемом носителе, например, водном носителе. Могут быть использованы различные водные носители, например, вода, буферная вода, 0,9% солевой раствор, 0,3% глицин, гиалуроновая кислота и тому подобное. Данные композиции можно стерилизовать обычными хорошо известными методами стерилизации, или их можно подвергать стерилизующей фильтрации. Полученные водные растворы могут быть упакованы для использования как есть, или лиофилизированы в виде лиофилизированного препарата, объединяемого со стерильным раствором перед введением. Композиции могут содержать фармацевтически приемлемые вспомогательные вещества, необходимые для аппроксимации физиологических условий, таких как агенты для регулирования уровня рН и буферные агенты, агенты, регулирующие тоничность, смачивающие агенты и тому подобное, например, ацетат натрия, лактат натрия, хлорид натрия, хлористый калий, хлорид кальция, сорбитанмонолаурат, олеат триэтаноламина и т.д.
[00234] Неоантигены могут также вводиться посредством липосом, которые нацеливают их на определенную ткань клеток, такую как лимфоидная ткань. Липосомы также полезны для увеличения периода полувыведения. Липосомы включают эмульсии, пены, мицеллы, нерастворимые монослои, жидкие кристаллы, фосфолипидные дисперсии, ламинарные слои и тому подобное. В этих препаратах неоантиген, который должен быть доставлен, включается как часть липосомы отдельно или в комбинации с молекулой, которая связывается, например, с рецептором, распространенным среди лимфоидных клеток, такими как моноклональные антитела, которые связываются с антигеном CD45, или с другими терапевтическими или иммуногенными композициями. Таким образом, липосомы, заполненные желаемым неоантигеном, могут быть направлены на сайт лимфоидных клеток, причем липосомы затем доставляют выбранные терапевтические/иммуногенные композиции. Липосомы могут быть образованы из стандартных везикулобразующих липидов, которые обычно содержат нейтральные и отрицательно заряженные фосфолипиды и стерол, такой как холестерин. Выбор липидов обычно осуществляется путем учета особенностей, например, размера липосом, кислотной лабильности и стабильности липосом в кровотоке. Для приготовления липосом доступны различные способы, как описано, например, Szoka et al., Ann. Rev. Biophys. Bioeng. 9; 467 (1980), патент США. №№4235871, 4501728, 4501728, 4837028, и 5019369.
[00235] Для нацеливания на иммунные клетки лиганд, который должен быть включен в липосому, может включать, например, антитела или их фрагменты, специфические для детерминант клеточной поверхности желаемых клеток иммунной системы. Липосомную суспензию можно вводить внутривенно, локально, местно и т.д. в дозе, которая варьируется в зависимости, в частности, от способа введения, доставки пептида и стадии заболевания, которое подлежит лечению.
[00236] Для терапевтических целей или целей иммунизации нуклеиновые кислоты, кодирующие пептид и необязательно один или более описанных в данном документе пептидов, также могут вводиться пациенту. Для доставки нуклеиновых кислот пациенту удобно использовать ряд способов. Например, нуклеиновая кислота может доставляться непосредственно, в «голой ДНК». Этот подход описан, например, в Wolff et al., Science 247: 1465-1468 (1990), а также в патенте США №№5580859 и 5589466. Нуклеиновые кислоты также можно вводить с использованием баллистической доставки, как описано, например, в патенте США №5204253. Частицы, состоящие исключительно из ДНК, можно вводить. Альтернативно, ДНК может быть прикреплена к частицам, таким как частицы золота. Подходы для доставки последовательностей нуклеиновых кислот могут включать вирусные векторы, векторы мРНК и векторы ДНК с или без электропорации.
[00237] Нуклеиновые кислоты также могут быть доставлены в комплексе с катионными соединениями, такими как катионные липиды. Способы липид-опосредованной доставки гена описаны, например, в 9618372 WO A WO 96/18372; 9324640 WO A WO 93/24640; Mannino & Gould-Fogerite, BioTechniques 6(7): 682-691 (1988); патенте США №5279833 Rose, патенте США №5279833; 9106309 WO A WO 91/06309; и Feigner et al., Proc. Natl. Acad. Sci. USA 84: 7413-7414(1987).
[00238] Неоантигены также могут быть включены в вакцинные платформы на основе вирусных векторных вакцин, таких как вирус осповакцины, вирус оспы кур, самовоспроизводящиеся альфавирусы, марабавирусы, аденовирусы (см., например, Tatsis et al., Adenoviruses, Molecular Therapy (2004) 10, 616-629), или лентивирус, включая, но не ограничиваясь этим, второй, третий или гибридный лентивирус второго и третьего поколений и рекомбинантный лентивирус любого поколения, предназначенный для нацеливания на конкретные типы клеток или рецепторы (см., например, Hu et al., Immunization Delivered by Lentiviral Vectors for Cancer and Infectious Diseases, Immunol Rev. (2011) 239(1): 45-61, Sakuma et al., Lentiviral vectors: basic to translational, Biochem J. (2012) 443(3):603-18, Cooper et al., Rescue of splicing-mediated intron loss maximizes expression in lentiviral vectors containing the human ubiquitin С promoter, Nucl. Acids Res. (2015) 43 (1): 682-690, Zufferey et al., Self-Inactivating Lentivirus Vector for Safe and Efficient In Vivo Gene Delivery, J. Virol. (1998) 72 (12): 9873-9880). В зависимости от упаковывающей способности вышеупомянутых вакцинных платформ на основе вирусных векторов данный подход может доставлять одну или более нуклеотидных последовательностей, которые кодируют один или более неоантигенных пептидов. Последовательности могут быть фланкированы немутированными последовательностями, могут быть разделены линкерами или им может предшествовать одна или более последовательностей, нацеленных на субклеточный компартмент (см., например, Gros et al., Prospective identification of neoantigen-specific lymphocytes in the peripheral blood of melanoma patients, Nat Med. (2016) 22 (4):433-8, Stronen et al., Targeting of cancer neoantigens with donor-derived T cell receptor repertoires, Science. (2016) 352 (6291):1337-41, Lu et al., Efficient identification of mutated cancer antigens recognized by T cells associated with durable tumor regressions, Clin Cancer Res. (2014) 20(13):3401-10). При введении в организм хозяина инфицированные клетки экспрессируют неоантигены и тем самым вызывают иммунную реакцию хозяина (например, ЦТЛ) против пептида(ов). Векторы на основе вируса осповакцины и способы, полезные в протоколах иммунизации, описаны, например, в патенте США №4722848. Другим вектором является BCG (бацилла Кальмета-Герена). Векторы на основе BCG описаны в Stover et al. (Nature 351:456-460 (1991)). Широкий спектр других векторов вакцины, полезных для терапевтического введения или иммунизации неоантигенов, например, векторов на основе Salmonella typhi, и тому подобное, будут очевидны специалистам в данной области техники из приведенного в данном документе описания.
[00239] Средство введения нуклеиновых кислот использует минигенные конструкции, кодирующие один или более эпитопов. Для создания последовательности ДНК, кодирующей выбранные ЦТЛ-эпитопы (миниген), для экспрессии в клетках человека, аминокислотные последовательности эпитопов обратно транслируют.Таблица частот использования кодонов человека используется для направления выбора кодонов для каждой аминокислоты. Эти последовательности ДНК, кодирующие эпитоп, непосредственно примыкают, создавая непрерывную полипептидную последовательность. Чтобы оптимизировать экспрессию и/или иммуногенность, конструкцию минигена могут быть включены дополнительные элементы. Примеры аминокислотной последовательности, которые могут быть обратно транслированы и включены в последовательность минигена, включают: хелперный Т-лимфоцит, эпитопы, лидерную (сигнальную) последовательность и сигнал удерживания в эндоплазматическом ретикулуме. Кроме того, ГКГС презентация ЦТЛ-эпитопов может быть улучшена путем включения синтетических (например, полиаланиновых) или встречающихся в природе фланкирующих последовательностей, смежных с ЦТЛ-эпитопами. Последовательность минигена преобразуют в ДНК путем сборки олигонуклеотидов, которые кодируют плюс- и минус- цепи минигена. Перекрывающие олигонуклеотиды (длиной 30-100 оснований) синтезируют, фосфорилируют, очищают и отжигают в подходящих условиях с использованием хорошо известных методов. Концы олигонуклеотидов соединяют с использованием ДНК-лигазы Т4. Данный синтетический миниген, кодирующий полипептид ЦТЛ-эпитопа, затем может быть клонирован в необходимый вектор экспрессии.
[00240] Очищенную плазмидную ДНК можно приготовить для инъекций с использованием различных составов. Простейшим из них является восстановление лиофилизированной ДНК в стерильном фосфатно-солевом буферном растворе (PBS). Были описаны различные методы, и новые методы могут стать доступными. Как отмечалось выше, нуклеиновые кислоты обычно получают с помощью катионных липидов. Кроме того, гликолипиды, фузогенные липосомы, пептиды и соединения совместно именуемые защитными, взаимодействующими, неконденсирующимися (PINC), также могут быть объединены в очищенную плазмидную ДНК, чтобы влиять на такие переменные, как стабильность, внутримышечная дисперсия или транспорт в определенные органы или типы клеток.
[00241] Также раскрыт способ изготовления противоопухолевой вакцины, включающий выполнение этапов способа, раскрытого в данном документе; и продуцирование противоопухолевой вакцины, содержащей множество неоантигенов или подсовокупность из совокупности неоантигенов.
[00242] Неоантигены, описанные в данном документе, могут быть изготовлены с использованием способов, известных в данной области техники. Например, способ получения неоантигена или вектора (например, вектор, содержащую по меньшей мере одну последовательность, кодирующую один или более неоантигенов), раскрытых в данном документе, может включать культивирование клетки-хозяина в условиях, подходящих для экспрессии неоантигена или вектора, причем клетка-хозяин содержит по меньшей мере один полинуклеотид, кодирующий неоантиген или вектор, и очистку неоантигена или вектора. Стандартные методы очистки включают хроматографические методы, электрофоретические, иммунологические методы, методы осаждения, диализа, фильтрации, концентрирования и хроматофокусирование.
[00243] Клетки-хозяева могут включать клетку яичника китайского хомяка (СНО), клетку NS0, дрожжи или клетку HEK293. Клетки-хозяева могут быть трансформированы одним или более полинуклеотидами, содержащими по меньшей мере одну последовательность нуклеиновой кислоты, которая кодирует неоантиген или вектор, раскрытый в данном документе, при этом, необязательно, выделенный полинуклеотид дополнительно содержит промоторную последовательность, функционально связанную с по меньшей мере одной последовательностью нуклеиновой кислоты, которая кодирует неоантиген или вектор. В некоторых вариантах осуществления выделенный полинуклеотид может представлять собой кДНК.
VI. Идентификация неоантигена
VI.A. Идентификация неоантигенного кандидата.
[00244] Методы исследований для NGS-анализа опухолей и нормальных экзома и транскриптомов были описаны и применены в отношении идентификации неоантигена.6,14,15 В приведенном ниже примере рассматриваются некоторые оптимизации для большей чувствительности и специфичности при идентификации неоантигена в клинических условиях. Эти оптимизации могут быть сгруппированы в две области, связанные с лабораторными процессами и те, которые связаны с анализом данных NGS.
VI.A.1. Оптимизация лабораторных процессов
[00245] Представленные в данном документе усовершенствования процесса устраняют проблемы в высокоточном обнаружении неоантигена из клинических образцов с низким содержанием и небольшими объемами опухолей, расширяя концепции, разработанные для надежной оценки генов рака, в целевых группах рака16, до установления полного экзома и транскриптома, необходимых для идентификации неоантигена. В частности, эти улучшения включают:
1. Нацеливание на глубокий (>500х) уникальный средний охват опухолевого экзома для выявления мутаций, присутствующих при низкой частоте встречаемости мутантного аллеля из-за низкого содержания опухоли или субклонального состояния.
2. Нацеливание на равномерный охват опухолевого экзома, пр и<5% оснований, покрытых<100х, благодаря чему пропущено наименьшее количество неоантигенов, например:
a. Применение зондов для захвата на основе ДНК с отдельным зондом QC17
b. Включение дополнительных приманок для слабоохваченных областей
3. Нацеливание на равномерный охват нормального экзома, где <5% оснований покрыты в <20х, так что наименьшее количество неоантигенов может оставаться неклассифицированными для статуса соматической/зародышевой линии (и, следовательно, не может использоваться в качестве TSNA)
4. Чтобы свести к минимуму общее количество требуемых секвенирований, зонды захвата последовательности будут предназначены только для кодирующих областей генов, поскольку некодирующая РНК не может привести к появлению неоантигенов.
Дополнительные оптимизации включают:
a. дополнительные зонды для генов HLA, которые богаты GC и слабозахвачены стандартным секвенированием экзома18
b. исключение генов, которые, как прогнозируется, генерируют мало или вообще не являются кандидатными неоантигенами, из-за таких факторов, как недостаточная экспрессия, субоптимальное расщепление протеасомами или необычные функции последовательности.
5. Опухолевая РНК также будет секвенирована при большой глубине (>100 прочтений), чтобы обеспечить возможность обнаружения вариантов, количественную оценку экспрессии гена и вариантов сплайсинга («изоформа») и обнаружение слияния. РНК из FFPE (зафиксированных в формалине и залитых парафином) образцов будет экстрагироваться с использованием обогащения на основе зондов19 с использованием тех же или аналогичных зондов, используемых для захвата экзомов в ДНК.
VI.A.2. Оптимизация анализа данных NGS
[00246] Усовершенствования методов анализа касаются субоптимальной чувствительности и специфичности общих исследовательских подходов определения мутаций, и, в частности, предусматривают настройки, необходимые для идентификации неоантигена в клинических условиях. В том числе:
1. Использование эталонного человеческого генома HG38 или более поздней версии для выравнивания, поскольку он включает сборки нескольких областей ГКГС, лучше отражающих полиморфизм популяции, в отличие от предыдущих релизов генома.
2. Преодоление ограничений определения отдельных вариантов20 путем объединения результатов из разных программ.5
a. Однонуклеотидные варианты и инсерционно-делеционные мутации будут обнаружены в опухолевой ДНК, опухолевой РНК и нормальной ДНК с помощью совокупности инструментов, в том числе: программ, основанных на сравнении опухолевой и нормальной ДНК, таких как Strelka21 и Mutect22; и программ, которые включают опухолевую ДНК, опухолевую РНК и нормальную ДНК, такую как UNCeqR, что особенно выгодно в образцах низкой чистоты23.
b. Инсерционно-делеционные мутации будут определяться с использованием программ, которые выполняют локальную повторную сборку, например, Strelka и ABRA24.
c. Структурные перестройки будут определяться с использованием специальных инструментов, таких как Pindel25 или Breakseq 26.
3. Чтобы выявлять и предотвращать замены образцов, определенные варианты из образцов для одного и того же пациента будут сравниваться с выбранным количеством полиморфных сайтов.
4. Обширная фильтрация артефактных определенных вариантов будет выполняться, например, путем:
a. Удаления вариантов, обнаруженных в нормальной ДНК, потенциально с ослабленными параметрами обнаружения в случаях низкого охвата, и с разрешающим критерием близости в случае инсерционно-делеционных мутаций
b. Удаления вариантов из-за низкого качества сопоставления или низкого базового качества27.
c. Удаления вариантов, возникающих из повторяющихся артефактов секвенирования, даже если они не наблюдается в соответствующем нормальном состоянии27. Примеры включают варианты, которые в первую очередь обнаруживаются на одной цепи.
d. Удаление вариантов, обнаруженных в несвязанной совокупности контролен27
5. Точное определение HLA из нормального экзома с использованием одного из seq2HLA28, ATHLATES29 или Optitype, а также комбинированных данных секвенирования экзома и РНК28. Дополнительные потенциальные оптимизации включают принятие специального анализа для HLA-типирования, такого как секвенирование для прочтения длинных ДНК30, или адаптация метода соединения фрагментов РНК с целью сохранения непрерывности 31.
6. Надежное обнаружение нео-ОРС, возникающих из опухолеспецифичеких сплайсированных вариантов, будет выполняться путем сборки транскриптов из данных РНК-секвенирования с использованием CLASS32, Bayesembler33, StringTie34 или аналогичной программы в режиме направленного сравнения (то есть с использованием известных структур транскрипции, а не попыток воссоздать транскрипты полностью из каждого эксперимента). В то время как Cufflinks35, обычно используемый для этой цели, часто приводит к невероятно большому числу сплайсированных вариантов, многие из которых намного короче, чем ген полной длины, и не могут восстановить простые положительные контроли. Кодирующие последовательности и потенциал нонсенс-опосредованного разрушения будут определяться с помощью таких инструментов, как SpliceR36 и МАМВА37, с повторными вставками мутантных последовательностей. Экспрессия генов будет определяться с помощью такого инструмента, как Cufflinks35 или Express (Roberts and Pachter, 2013). Количество и/или относительных уровней экспрессии у дикого типа и мутантов будут определяться с помощью инструментов, разработанных для этих целей, таких как ASE38 или HTSeq39. Потенциальные этапы фильтрации включают:
a. Удаление кандидатных нео-ОРС, которые считаются недостаточно экспрессируемыми.
b. Удаление кандидатных нео-ОРС, спрогнозированных для запуска нонсенс-опосредованного разрушения (NMD).
7. Кандидатные неоантигены, наблюдаемые только в РНК (например, нео-ОРС), которые не могут быть непосредственно подтверждены как специфические к опухоли, будут классифицированы как вероятно специфические к опухоли в соответствии с дополнительными параметрами, например, с учетом:
a. Наличия поддерживающей опухолевую ДНК только цис-действующих мутаций со сдвигом рамки считывания или сайтов сплайсинга.
b. Наличия подтвержденной опухолевой ДНК только транс-действующей мутации в факторе сплайсинга. Например, в трех независимо опубликованных экспериментах с R625-мутантом SF3B1 гены, проявляющие наиболее дифференцированный сплайсинг, были согласующимися, хотя в одном эксперименте рассматривались пациенты с увеальной меланомой40, во втором - линия клеток увеальной меланомы41 и в третьем - пациенты с раком молочной железы 42.
с.Для новых сплайсинговых изоформ присутствие подтверждающего «нового» экзон-интронное сочленения читается в данных РНК-секвенирования.
d. Для новых перестроек присутствие подтверждающих рядом экзонов считывается в опухолевой ДНК, которые отсутствуют в нормальной ДНК
e. Отсутствия в перечне экспрессии генов, такого как GTEx43 (т.е. что делает возникновение зародышевой линии менее вероятным)
8. В дополнение к анализу, основанному на выравнивании эталонного генома, путем сравнения собранных ДНК опухолей и нормальных чтений (или k-меров из таких чтений), чтобы напрямую избежать ошибок и артефактов на основе выравнивания и аннотации, (например, для соматических вариантов, возникающих вблизи вариантов зародышевой линии или повторяющихся контекстных инсерционно-делеционных мутаций)
[00247] В образцах с полиаденилированной РНК присутствие вирусной и микробной РНК в данных РНК-секвенирования будет оцениваться с использованием RNA CoMPASS44 или аналогичного метода для идентификации дополнительных факторов, которые могут прогнозировать реакцию пациента.
VI.B Выделение и обнаружение пептидов HLA
[00248] Выделение молекул HLA-пептида проводили с использованием классических методов иммунопреципитации (ИП) после лизиса и солюбилизации образца ткани (55-58). Осветленный лизат использовали для HLA-специфической ИП.
[00249] Иммунопреципитацию проводили с использованием антител, связанных с гранулами, при этом антитело было специфичным для молекул HLA. Для иммунопреципитации HLA пан-класса I используется антитело к CR пан-класса I, для HLA-DR класса II используется антитело к HLA-DR. Антитело ковалентно прикрепляют к NHS-сефарозным гранулам (NHS-N-гидроксисукцинимид) во время инкубации в течение ночи. После ковалентного прикрепления гранулы промывали и аликвотировали для ИП. (59, 60)
[00250] Осветленный тканевый лизат добавляют к нагруженным антителам гранулам для иммунопреципитации. После иммунопреципитации гранулы удаляют из лизата и лизат хранят для дополнительных экспериментов, включая дополнительные ИП. Гранулы для ИП промывают для удаления неспецифического связывания и комплекс HLA/пептид элюируют из гранул стандартными методами. Белковые компоненты удаляют из пептидов с использованием молекулярновесовой спин-колонки или фракционирования С18. Полученные в результате пептиды высушивают путем выпаривания SpeedVac и в некоторых случаях хранят при -20°С до анализа МС.
[00251] Высушенные пептиды восстанавливают в буфере для ВЭЖХ, подходящем для хроматографии с обращенной фазой, и загружают в микрокапиллярную хроматографическую колонку С-18 для градиентного элюирования в масс-спектрометре Fusion Lumos (Thermo). Спектры MSI массы/заряда пептида (масса/заряда) собирали в детекторе Orbitrap с высоким разрешением с последующим сканированием MS2 с низким разрешением, собранным в детекторе с ионной ловушкой после фрагментации HCD выбранного иона. Кроме того, спектры MS2 могут быть получены с использованием методов фрагментации CID или ETD или любой комбинации этих трех методов для достижения большего покрытия аминокислотами пептида. Спектры MS2 также могут быть измерены с большой разрешающей способностью массы в детекторе Orbitrap.
[00252] Спектры MS2 от каждого анализа исследуются с помощью базы данных по структуре белка с использованием Comet (61, 62), а идентификацию пептида оценивают с использованием Percolator (63-65).
VT.B.1. Исследования предела обнаружения МС для обеспечения выполнения полного секвенирования пептида HLA.
[00253] С использованием пептида YVYVADVAAK было определено, какие пределы обнаружения используют разные количества пептида, загруженного в колонку для ЖХ. Количество исследуемого пептида составляло 1 пмоль, 100 ммоль, 10 фмоль, 1 фмоль и 100 амоль. (Таблица 1) Результаты изображены на фигуре 1F. Эти результаты демонстрируют, что нижний предел обнаружения (LoD) находится в аттомолярном диапазоне (10-18), что динамический диапазон охватывает пять порядков и что сигнал к шуму является достаточным для секвенирования в низких фемтомолярных диапазонах (10-15).
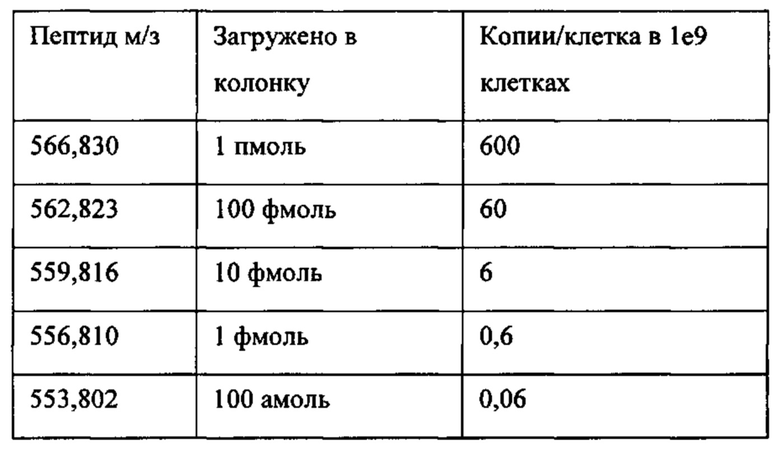
VII. Модель презентации
VII.A. Системный обзор
[00254] На фиг. 2А представлен обзор среды 100 для определения вероятности презентации пептида у пациентов в соответствии с вариантом осуществления. Среда 100 обеспечивает контекст для введения системы идентификации презентации 160, которая содержит хранилище информации о презентации 165.
[00255] Система идентификации презентации 160 представляет собой одну или компьютерные модели, воплощенные в вычислительной системе, как обсуждается ниже со ссылкой на фиг. 14, который принимает пептидные последовательности, связанные с совокупностью аллелей ГКГС, и определяет вероятность того, что пептидные последовательности будут презентированы одним или более совокупностями ГКГС-ассоциированных аллелей. Это полезно в различных контекстах. Один конкретный вариант использования для системы идентификации презентации 160 состоит в том, что она способна принимать нуклеотидные последовательности кандидатные неоантигенов, связанных с совокупностью аллелей ГКГС из опухолевых клеток пациента 110, и определять вероятность того, что кандидатные неоантигены будут презентированы одним или более ГКГС-ассоциированными аллелями опухоли, и/или индуцировать иммуногенные ответы в иммунной системе пациента 110. Те кандидатные неоантигены с высокой вероятностью, которые определены при помощи системы 160, могут быть выбраны для включения в вакцину 118, такой противоопухолевый иммунный ответ может быть вызван иммунной системой пациента 110, обеспечивающего опухолевые клетки.
[00256] Система идентификации презентации 160 определяет вероятности презентации посредством одной или более моделей презентации. В частности, модели презентации генерируют вероятность того, будут ли данные пептидные последовательности презентированы совокупностью ГКГС-ассоциированных аллелей и сформированы на основе информации о презентации, хранимой в хранилище 165. Например, модели презентации могут генерировать вероятность того, будет ли презентирована пептидная последовательность «YVYVADVAAK» для совокупности аллелей HLA-A*02:01, HLA-В*07:02, HLA-B*08:03, HLA-C*01:04, HLA-A*06:03, HLA-B*01:04 на поверхности клетки образца. Информация о презентации 165 содержит информацию о том, связываются ли пептиды с различными типами аллелей ГКГС, так что эти пептиды презентированы аллелями ГКГС, которые в моделях определяются в зависимости от положения аминокислот в пептидных последовательностях. Модель презентации может спрогнозировать, будет ли нераспознанная пептидная последовательность презентирована вместе с ассоциированной совокупностью аллелей ГКГС на основе информации о презентации 165.
VII.В. Информация о презентации
[00257] На фиг. 2 изображен способ получения информации презентации в соответствии с вариантом осуществления. Информация о презентации 165 содержит две общие категории информации: аллель-взаимодействующую информацию и аллель-невзаимодействующую информацию. Аллель-взаимодействующая информация содержит информацию, которая влияет на презентацию пептидных последовательностей, которые зависят от типа аллеля ГКГС.Аллель-невзаимодействующая информация содержит информацию, которая влияет на презентацию пептидных последовательностей, которые не зависят от типа аллеля ГКГС.
VII.B.1. Аллель-взаимодействующая информация
[00258] Аллель-взаимодействующая информация в основном включает идентифицированные пептидные последовательности, которые, как известно, были презентированы одной или более идентифицированными молекулами ГКГС от людей, мышей и т.д. Примечательно, что она может включать или не включать данные, полученные из образцов опухоли. Презентированные пептидные последовательности могут быть идентифицированы из клеток, которые экспрессируют единичную аллель ГКГС. В этом случае презентированные пептидные последовательности обычно собирают из одноаллельных клеточных линий, которые модифицированы для экспрессии заданного аллеля ГКГС и которые затем подвергаются воздействию синтетического белка. Пептиды, презентированные на аллеле ГКГС, выделяют с помощью таких методов, как кислотное элюирование и идентифицируют с помощью масс-спектрометрии. На фиг. 2 В изображен пример вышеуказанного, в котором иллюстративный пептид YEMFNDKS, презентирован на предопределенном аллеле ГКГС HLA-A*01:01, выделяют и идентифицируют с помощью масс-спектрометрии. Так как в этой ситуации пептиды идентифицированы с помощью клеток, модифицированных для экспрессии одного предопределенного белка ГКГС, то прямая связь между презентированным пептидом и белком ГКГС, с которым он был связан, определенно известна.
[00259] Презентированные пептидные последовательности также могут быть собраны из клеток, которые экспрессируют множественные аллели ГКГС. Обычно у людей для клетки экспрессируются 6 различных типов молекул ГКГС. Такие презентированные пептидные последовательности могут быть идентифицированы из многоаллельных клеточных линий, которые модифицированы для экспрессии нескольких предопределенных аллелей ГКГС. Такие презентированные пептидные последовательности также могут быть идентифицированы из образцов ткани или из образцов нормальной ткани, или из образцов опухолевой ткани. В этом случае, в частности, молекулы ГКГС могут быть иммунопреципитированы из нормальной или опухолевой ткани. Пептиды, презентированные на множественных аллелях ГКГС, можно аналогичным образом выделять с помощью таких методов, как кислотное элюирование и идентифицировать с помощью масс-спектрометрии. На фиг. 2С изображен пример вышеуказанного, в котором шесть идентифицированных пептидов YEMFNDKSF, HROEIFSHDFJ, FJIEJFOESS, NEIOREIREI, JFKSIFEMMSJDSSU и KNFLENFIESOFI, презентированные на идентифицированных аллелях ГКГС HLA-A*01:01, HLA-A*02:01, HLA-B*07:02, HLA-B*08: 01, HLA-C*01:03 и HLA-C*01:04, и выделяют и идентифицируют с помощью масс-спектрометрии. В отличие от одноаллельных клеточных линий, прямая связь между презентированным пептидом и белком ГКГС, с которым он был связан, может быть неизвестным, поскольку связанные пептиды выделены из молекул ГКГС до их идентификации.
[00260] Аллель-взаимодействующая информация может также включать ионный ток масс-спектрометрии, который зависит как от концентрации комплексов пептид-молекула ГКГС, так и от эффективности ионизации пептидов. Эффективность ионизации варьируется от пептида к пептиду зависимым от последовательности образом. Как правило, эффективность ионизации варьируется от пептида к пептиду в пределах приблизительно двух порядков, тогда как концентрация комплексов пептид-ГКГС колеблется в более широком диапазоне, чем это.
[00261] Аллель-взаимодействующая информация может также включать измерения или прогнозирования аффинности связывания между данным аллелем ГКГС и данным пептидом. Одна или более моделей аффинности могут генерировать такие прогнозы. Например, возвращаясь к примеру, изображенному на фиг. 1D, информация о презентации 165 может включать прогнозирование аффинности связывания в 1000 нМ между пептидом YEMFNDKSF и аллелем HLA-A*01:01. Несколько пептидов с ИК50 >1000 нм ГКГС презентировано, а более низкие значения ИК50 увеличивают вероятность презентации.
[00262] Аллель-взаимодействующая информация может также включать измерения или прогнозы стабильности комплекса ГКГС. Одна или более моделей стабильности, которые могут генерировать такие прогнозы. Более стабильные комплексы пептид-МНС (то есть комплексы с более длительным периодом полувыведения), скорее всего, будут презентированы с большим числом копий на опухолевых клетках и на антигенпрезентирущих клетках, которые сталкиваются с вакцинным антигеном. Например, возвращаясь к примеру, изображенному на фиг. 2С, информация о презентации 165 может включать прогнозирование стабильности периода полувыведения в 1 ч для молекулы HLA-А*00:01.
[00263] Аллель-взаимодействующая информация может также включать измеренную или прогнозируемую скорость реакции образования комплекса пептид-ГКГС. Комплексы, которые формируются с более высокой скоростью, с большей вероятностью будут презентированы на поверхности клетки в высокой концентрации.
[00264] Аллель-взаимодействующая информация может также включать последовательность и длину пептида. Молекулы ГКГС класса I обычно предпочитают презентировать пептиды с длиной от 8 до 15 пептидов. 60-80% презентированных пептидов имеют длину 9. Гистограммы представленных пептидных длин из нескольких линий клеток изображены на фиг. 5.
[00265] Аллель-взаимодействующая информация может также включать присутствие киназных мотивов последовательности на кодируемом неоантигеном пептиде и отсутствие или наличие специфических посттрансляционных модификаций в кодируемом неоантигеном пептиде. Наличие киназных мотивов влияет на вероятность посттрансляционной модификации, которая может усиливать или мешать связыванию ГКГС.
[00266] Аллель-взаимодействующая информация может также включать уровни экспрессии или активности белков, участвующих в процессе посттрансляционной модификации, например, киназы (измеренные или спрогнозированные с использованием РНК-секвенирования, масс-спектрометрии или других методов).
[00267] Аллель-взаимодействующая информация также может включать вероятность презентации пептидов с аналогичной последовательностью в клетках от других индивидуумов, экспрессирующих специфический аллель ГКГС, как оценивается с помощью протеомного масс-спектрометрического анализа или другими способами.
[00268] Аллель-взаимодействующая информация может также включать уровни экспрессии конкретного аллеля ГКГС у индивидуума, о котором идет речь (например, как измерено с помощью РНК-секвенирования или масс-спектрометрии). Пептиды, которые наиболее сильно связываются с аллелем ГКГС, который экспрессируется на высоких уровнях, скорее всего будут презентированы, в отличие от пептидов, которые наиболее сильно связываются с аллелем ГКГС, который экспрессируется на низком уровне.
[00269] Аллель-взаимодействующая информация также может включать независимую вероятность презентации кодируемой неоантигеном пептидной последовательности конкретным аллелем ГКГС у других индивидуумов, которые экспрессируют конкретный аллель ГКГС.
[00270] Аллель-взаимодействующая информация может также включать независимую вероятность презентации полной пептидной последовательности аллелями ГКГС в одном и том же семействе молекул (например, HLA-A, HLA-B, HLA-C, HLA-DQ, HLA-DR, HLA-DP) у других индивидуумов. Например, молекулы HLA-C обычно экспрессируются на более низких уровнях, чем молекулы HLA-A или HLA-B, и, следовательно, презентация пептида HLA-C является априори менее вероятным, чем презентация HLA-A или HLA-B11.
[00271] Аллель-взаимодействующая информация может также включать белковую последовательность конкретного аллеля ГКГС.
[00272] Любая аллель-невзаимодействующая информация ГКГС, указанная в следующем разделе, также может быть смоделирована как ГКГС-аллель-связывающая информация.
VII.B.2. Аллель-невзаимодействующая информация
[00273] Аллель-невзаимодействующая информация может включать С-концевые последовательности, фланкирующие кодируемый неоантигеном пептид в его исходной белковой последовательности. С-концевые фланкирующие последовательности могут влиять на протеасомный процессинг пептидов. Однако С-концевая фланкирующая последовательность отщепляется от пептида при помощи протеасомы до того, как пептид транспортируется в эндоплазматический ретикулум и встречается с аллелями ГКГС на поверхностях клеток. Следовательно, молекулы ГКГС не получают никакой информации о С-концевой фланкирующей последовательности, и, следовательно, влияние С-концевой фланкирующей последовательности не может меняться в зависимости от типа аллелей ГКГС. Например, возвращаясь к примеру, изображенному на фиг. 2С, информация о презентации 165 может включать С-концевую фланкирующую последовательность FOEIFNDKSLDKFJI презентированного пептида FJIEJFOESS, идентифицированного из исходного белка пептида.
[00274] Аллель-невзаимодействующая информация может также включать количественные измерения мРНК. Например, данные количественной оценки мРНК могут быть получены для тех же образцов, которые обеспечивают масс-спектрометрические данные для обучения. Как более подробно описано со ссылкой на фиг. 13Н, было определено, что экспрессия РНК является сильным прогностическим фактором презентации пептидов. В одном варианте осуществления количественные измерения мРНК идентифицируют из инструментального средства программного обеспечения RSEM. Подробную реализацию программного обеспечения RSEM можно найти в Во Li и Colin N. Dewey. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC Bioinformatics, 12:323, August 2011. В одном варианте осуществления количественную оценку мРНК отображают в единицах фрагментов на килобазу транскрипта на миллион картированных прочтений (FPKM).
[00275] Аллель-невзаимодействующая информация может также включать N-концевые последовательности, фланкирующие пептид в его исходной белковой последовательности.
[00276] Аллель-невзаимодействующая информация может также включать присутствие мотивов расщепления протеазой в пептиде, необязательно оцененного в соответствии с экспрессией соответствующих протеаз в опухолевых клетках (как измерено с помощью РНК-секвенирования или масс-спектрометрии). Пептиды, которые содержат мотивы расщепления протеазой, с меньшей вероятностью будут презентированы, потому что они будут более быстро деградированы протеазами и поэтому будут менее стабильными в клетке.
[00277] Аллель-невзаимодействующая информация может также включать интенсивность обмена исходного белка, измеренную в соответствующем типе клеток. Более высокая интенсивность обмена (т.е. более низкий период полувыведения) увеличивает вероятность презентации; однако прогностическая мощность этого свойства низкая, если измеряется в разнородном типе клеток.
[00278] Аллель-невзаимодействующая информация может также включать длину исходного белка, необязательно учитывая конкретные варианты сплайсинга («изоформы»), наиболее высоко экспрессируемые в опухолевых клетках, как измерено с помощью РНК-секвенирования или масс-спектрометрии протеома, или как спрогнозировано из аннотации сплайсинговых мутаций соматических или зародышевой линии, обнаруженных в данных последовательностей ДНК или РНК.
[00279] Аллель-невзаимодействующая информация также может включать уровень экспрессии протеасомы, иммунопротеасомы, тимопротеасомы или других протеаз в опухолевых клетках (которые могут быть измерены с помощью РНК-секвенирования, масс-спектрометрии протеома или иммуногистохимии). Различные протеасомы имеют разные предпочтения к сайту расщепления. Больший вес будет отдаваться предпочтениям расщепления каждого типа протеасомы пропорционально ее уровню экспрессии.
[00280] Аллель-невзаимодействующая информация может также включать экспрессию исходного гена пептида (например, как измерено с помощью РНК-секвенирования или масс-спектрометрии). Возможные оптимизации включают корректировку измеренной экспрессии для учета наличия стромальных клеток и инфильтрирующих опухоли лимфоцитов в образце опухоли. Вероятнее всего, будут презентированы пептиды из более экспрессированных генов. Пептиды из генов с неопределяемыми уровнями экспрессии могут быть исключены из рассмотрения.
[00281] Аллель-невзаимодействующая информация также может включать вероятность того, что исходная мРНК кодируемого неоантигеном пептида будет подвергнута нонсенс-опосредованному разрушению, как спрогнозировано при помощи модели нонсенс-опосредованного разрушения, например, модель из Rivas et al, Science 2015.
[00282] Аллель-невзаимодействующая информация может также включать типичную тканеспецифическую экспрессию исходного гена пептида на разных стадиях клеточного цикла. Гены, которые экспрессируются на низком уровне в целом (как измерено РНК-секвенированием или протеомным масс-спектрометрическим анализом), но которые, как известно, экспрессируются на высоком уровне на определенных стадиях клеточного цикла, скорее всего, будут продуцировать более презентированные пептиды, чем гены, которые стабильно экспрессируются на очень низких уровнях.
[00283] Аллель-невзаимодействующая информация может также включать полный каталог характеристик исходного белка, как указано в, например, uniProt или PDB http://www.rcsb.org/pdb/home/home.do. Эти характеристики могут включать, среди прочего, вторичную и третичную структуры белка, субклеточную локализацию 11, термины генной онтологии (GO). В частности, эта информация может включать аннотации, которые действуют на уровне белка, например, длину 5''-НТО, и аннотации, которые действуют на уровне конкретных остатков, например, спиральный мотив между остатками 300 и 310. Эти признаки также могут включать мотивы поворота, мотивы складчатости и неупорядоченные остатки.
[00284] Аллель-невзаимодействующая информация может также включать признаки, описывающие свойства домена исходного белка, содержащего пептид, например: вторичную или третичную структуру (например, альфа-спираль против бета-листа); альтернативный сплайсинг
[00285] Аллель-невзаимодействующая информация может также включать признаки, описывающие наличие или отсутствие гипервариабельного участка при презентации в положении пептида в исходном белке пептида.
[00286] Аллель-невзаимодействующая информация также может включать вероятность презентации пептидов из исходного белка рассматриваемого пептида у других индивидуумов (после корректировки уровня экспрессии исходного белка у этих индивидуумов и влияния различных типов HLA этих индивидуумов).
[00287] Аллель-невзаимодействующая информация может также включать вероятность того, что пептид не будет обнаружен или сверхпрезентирован при помощи масс-спектрометрии из-за технических ошибок.
[00288] Экспрессия различных генных модулей/путей, измеренных с помощью анализа экспрессии гена, такого как РНК-секвенирование, микрочипы(ы), целевые панели(ы), такие как Nanostring, или олигонуклеотидные представители генных модулей, измеренные с помощью анализов, таких как ОТ-ПЦР (которые не обязательно содержат исходный белок пептида), которые являются информативными относительно состояния опухолевых клеток, стромы или инфильтрирующих опухоли лимфоцитов (TIL).
[00289] Аллель-невзаимодействующая информация также может включать число копий исходного гена пептида в опухолевых клетках. Например, пептидам из генов, которые подвержены гомозиготной делеции в опухолевых клетках, может быть назначена вероятность презентации равная нулю.
[00290] Аллель-невзаимодействующая информация также может включать вероятность того, что пептид связывается с ТАР, или измеренную или спрогнозированную аффинность связывания пептида с ТАР. Вероятнее всего, будут презентированы пептиды, которые более склонны связываться с ТАР, или пептиды, которые связывают ТАР с более высокой аффинностью.
[00291] Аллель-невзаимодействующая информация может также включать уровень экспрессии ТАР в опухолевых клетках (который может быть измерен с помощью РНК-секвенирования, масс-спектрометрии протеома, иммуногистохимии). Более высокие уровни экспрессии ТАР повышают вероятность презентации всех пептидов.
[00292] Аллель-невзаимодействующая информация может также включать наличие или отсутствие опухолевых мутаций, включая, но не ограничиваясь ими:
i. Драйверные мутации в известных генах-драйверах рака, такие как EGFR, KRAS, ALK, RET, ROS1, ТР53, CDKN2A, CDKN2B, NTRK1, NTRK2, NTRK3
ii. В генах, кодирующих белки, участвующие в механизмах презентации антигенов (например, В2М, HLA-A, HLA-B, HLA-C, ТАР-1, ТАР-2, ТАРВР, CALR, CNX, ERP57, HLA-DM, HLA-DMA, HLA-DMB, HLA-DO, HLA-DOA, HLA-DOBHLA-DP, HLA-DPA1, HLA-DPB1, HLA-DQ, HLA-DQA1, HLA-DQA2, HLA-DQB1, HLA-DQB2, HLA-DR, HLA-DRA, HLA-DRB1, HLA-DRB3, HLA-DRB4, HLA-DRB5 или любом из генов, кодирующих компоненты протеасомы или иммунопротеасомы). Пептиды, чья презентация опирается на компонент механизма презентации антигена, который подвержен мутации с утратой функции в опухоли, снизили вероятность презентации.
[00293] Наличие или отсутствие функциональных полиморфизмов зародышевой линии, включая, но не ограничиваясь:
i. В генах, кодирующих белки, участвующие в механизмах презентации антигенов (например, В2М, HLA-A, HLA-B, HLA-C, ТАР-1, ТАР-2, ТАРВР, CALR, CNX, ERP57, HLA-DM, HLA-DMA, HLA-DMB, HLA-DO, HLA-DOA, HLA-DOBHLA-DP, HLA-DPA1, HLA-DPB1, HLA-DQ, HLA-DQA1, HLA-DQA2, HLA-DQB1, HLA-DQB2, HLA-DR, HLA-DRA, HLA-DRB1, HLA-DRB3, HLA-DRB4, HLA-DRB5 или любом из генов, кодирующих компоненты протеасомы или иммунопротеасомы).
[00294] Аллель-невзаимодействующая информация может также включать тип опухоли (например, НМРЛ, меланома).
[00295] Аллель-невзаимодействующая информация может также включать известную функциональность аллелей HLA, что отражается, например, на суффиксах аллелей HLA. Например, суффикс N в названии аллели HLA-A*24: 09N указывает на молчащий аллель, который не экспрессруется и поэтому вряд ли будет презентировать эпитопы; полная номенклатура суффиксов аллелей HLA описана в https://www.ebi.ac.uk/ipd/imgt/hla/nomenclature/suffixes.html.
[00296] Аллель-невзаимодействующая информация может также включать клинический подтип опухоли (например, плоскоклеточный рак легкого в противовес неплоскоклеточного).
[00297] Аллель-невзаимодействующая информация также может включать анамнез курения.
[00298] Аллель-невзаимодействующая информация может также включать историю солнечных ожогов, ультрафиолетовое облучение или воздействие других мутагенов.
[00299] Аллель-невзаимодействующая информация может также включать типичную экспрессию исходного гена пептида в соответствующем типе опухоли или клиническом подтипе, необязательно стратифицированном при помощи драйверной мутации. Гены, которые обычно экспрессируются на высоких уровнях в соответствующем типе опухоли, скорее всего, будут презентированы.
[00300] Аллель-невзаимодействующая информация может также включать частоту мутации во всех опухолях или опухолях того же типа или в опухолях у людей с по меньшей мере одним общим аллелем ГКГС или опухолями того же типа у индивидуумов с по меньшей мере одним общим аллелем ГКГС.
[00301] В случае мутированного опухолеспецифического пептида список признаков, используемых для прогнозирования вероятности презентации, может также включать аннотацию мутации (например, миссенс, сквозного прочитывания, со сдигом рамки считывания, слития и т.д.) или же мутация как спрогнозировано, приведет к нонсенс-опосредованному разрушению (NMD). Например, пептидам из белковых сегментов, которые не транслированы в опухолевых клетках из-за гомозиготных мутаций с преждевременным стоп-кодоном, могут быть назначены вероятности презентации равные нулю. NMD приводит к снижению трансляции мРНК, что снижает вероятность презентации.
VTI.C. Система идентификации презентации
[00302] На фиг. 3 представлена высокоуровневая блок-схема, изображающая компоненты компьютерной логики системы идентификации презентации 160, в соответствии с одним вариантов осуществления. В данном иллюстративном варианте осуществления система идентификации презентации 160 включает модуль управления данными 312, кодирующий модуль 314, обучающий модуль 316 и модуль прогнозирования 320. Система идентификации презентации 160 также включает хранилище данных для обучения 170 и хранилище моделей презентации 175. Некоторые варианты осуществления системы управления моделями 160 имеют разные модули, отличные от описанных в данном документе. Аналогично, функции могут быть распределены между модулями по-другому, чем описано в данном документе.
VII.C.1. Модуль управления данными
[00303] Модуль управления данными 312 генерирует совокупности данных для обучения 170 из информации о презентации 165. Каждая совокупность данных для обучения содержит множество объектов данных, в которых объект данных i содержит совокупность независимых переменных zi, которые включают по меньшей мере презентированную или непрезентированную пептидную последовательность pi, один или более ГКГС-ассоциированных аллелей ai, связанных с пептидной последовательностью pi и зависимую переменную yi, которая представляет информацию, которую система идентификации презентации 160 использует в прогнозировании новых значений независимых переменных.
[00304] В одном конкретном варианте осуществления, упомянутом в оставшейся части описания, зависимая переменная yi является двоичной меткой, указывающей, был ли пептид pi презентирован одним или более ГКГС-ассоциированными аллелями ai. Однако следует понимать, что в других реализациях зависимая переменная yi может представлять любую другую информацию, которую система идентификации презентации 160 использует в прогнозировании, зависящем от независимых переменных zi. Например, в другой реализации зависимая переменная yi также может быть числовой величиной, указывающей на масс-спектрометрический ионный ток, идентифицированный для объекта данных.
[00305] Пептидная последовательность pi для элемента данных i представляет собой последовательность ki аминокислот, в которой ki может варьироваться между объектами данных i в пределах диапазона. Например, этот диапазон может составлять 8-15 для ГКГС класса I или 9-30 для ГКГС класса II. В одной конкретной реализации системы 160 все пептидные последовательности pi в совокупности данных для обучения могут иметь одинаковую длину, например, 9. Количество аминокислот в пептидной последовательности может изменяться в зависимости от типа аллелей ГКГС (например, аллели ГКГС у людей и т.д.). Аллели ГКГС ai для объекта данных i указывают, какие аллели ГКГС присутствуют в связи с соответствующей пептидной последовательностью pi.
[00306] Модуль управления данными 312 также может включать дополнительные аллель-взаимодействующие переменные, такие как прогнозы аффинности связывания bi и стабильности si в сочетании с пептидными последовательностями pi и ГКГС-ассоциированными аллелями ai, содержащимися в данных для обучения 170. Например, данные для обучения 170 могут включать прогнозы аффинности связывания bi между пептидом pi и каждой из ГКГС-ассоциированых молекул, указанных в ai. В качестве другого примера данные для обучения 170 могут включать прогнозы стабильности si для каждого из аллелей ГКГС, указанных ai.
[00307] Модуль управления данными 312 также может включать аллель-невзаимодействующие переменные wi, такие как С-концевые фланкирующие последовательности и количественные измерения мРНК в сочетании с пептидными последовательностями pi.
[00308] Модуль управления данными 312 также идентифицирует пептидные последовательности, которые не презентированы аллелями ГКГС, чтобы генерировать данные для обучения 170. Как правило, он включает идентификацию «более длинных» последовательностей исходного белка, которые содержат презентированные пептидные последовательности перед их презентацией. Когда информация о презентации включает модифицированные линии клеток, модуль управления данными 312 идентифицирует серию пептидных последовательностей, которые не были презентированы на аллелях ГКГС клеток, в синтетическом белке, воздействию которого подвергались клетки. Когда информация о презентации включает образцы ткани, модуль управления данными 312 идентифицирует исходные белки, из которых происходят презентированные пептидные последовательности, и идентифицирует серию пептидных последовательностей в исходном белке, которые не были презентированы на аллелях ГКГС клеток образца ткани.
[00309] Модуль управления данными 312 также может искусственно генерировать пептиды со случайными последовательностями аминокислот и идентифицировать генерируемые последовательности в виде пептидов, не презентированных на аллелях ГКГС. Это может быть выполнено путем случайного генерирования пептидных последовательностей, позволяющих модулю управления данными 312 легко генерировать большое количество синтетических данных для пептидов, не презентированных на аллелях ГКГС. Поскольку на самом деле небольшой процент пептидных последовательностей презентирован аллелями ГКГС, синтетически генерируемые пептидные последовательности, скорее всего, не были бы презентированы аллелями ГКГС, даже если они были включены в белки, процессируемые клетками.
[00310] На фиг. 4 изображен иллюстративная совокупность данных для обучения 170А в соответствии с одним вариантом осуществления. В частности, первые 3 объекта данных в данных для обучения 170А указывают на информацию о презентации пептида из одноаллельной клеточной линии, включая аллельные последовательности HLA-C*01:03 и 3 пептидные последовательности QCEIOWARE, FIEUHFWI и FEWRHRJTRUJR. Четвертый объект данных в данных для обучения 170А указывает на информацию о пептиде из многоаллельной клеточной линии, включая аллели HLA-B*07:02, HLA-C*01:03, HLA-A*01:01 и пептидную последовательность QIEJOEIJE. Первый объект данных указывает на то, что пептидная последовательность QCEIOWARE не была презентирована аллелем HLA-C*01:03. Как обсуждалось в предыдущих двух параграфах, последовательность пептидов может быть случайно сгенерирована модулем управления данными 312 или идентифицирована из исходного белка презентированных пептидов. Данные для обучения 170А также включают прогнозирование аффинности связывания 1000 нМ и прогнозирование стабильности периода полувыведения в течение 1 часа для пары пептидная последовательность-аллель. Данные для обучения 170А также включают аллель-невзаимодействующие переменные, такие как С-концевая фланкирующая последовательность пептида FJELFISBOSJFIE и количественное измерение мРНК 102 FPKM. Четвертый объект данных указывает на то, что пептидная последовательность QIEJOEIJE была презентирована одним из аллелей HLA-B*07:02, HLA-C*01:03, или HLA-А*01:01. Данные для обучения 170А также включают прогнозы аффинности связывания и прогнозы стабильности для каждого из аллелей, а также С-фланкирующую последовательность пептида и количественное измерение мРНК для пептида.
VII.C.2. Кодирующий модуль
[00311] Кодирующий модуль 314 кодирует информацию, содержащуюся в данных для обучения 170, в числовая презентация, которая может использоваться для создания одной или более моделей презентации. В одном варианте осуществления кондирующий модуль 314 прямо унитарно кодирует последовательности (например, пептидные последовательности или С-концевые фланкирующие последовательности) по заранее заданному 20-буквенному аминокислотному алфавиту. В частности, пептидная последовательность pi с ki аминокислотами представлена в виде вектор-ряда из 20⋅ki элементов, причем один элемент среди 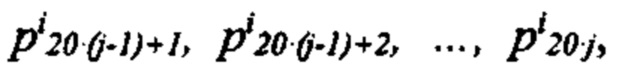 что соответствует алфавиту аминокислоты в j-м положении пептидной последовательности, имеет значение 1. В противном случае остальные элементы имеют значение 0. В качестве примера, для данного алфавита {А, С, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W, Y}, пептидная последовательность EAF из 3 аминокислот для объекта данных i может быть представлена вектором строк из 60 элементов pi=[0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0]. С-концевую фланкирующую последовательность ci можно кодировать так же, как описано выше, а также последовательность белка dh для аллелей ГКГС и другие данные последовательности в информации о презентации.
что соответствует алфавиту аминокислоты в j-м положении пептидной последовательности, имеет значение 1. В противном случае остальные элементы имеют значение 0. В качестве примера, для данного алфавита {А, С, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W, Y}, пептидная последовательность EAF из 3 аминокислот для объекта данных i может быть представлена вектором строк из 60 элементов pi=[0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0]. С-концевую фланкирующую последовательность ci можно кодировать так же, как описано выше, а также последовательность белка dh для аллелей ГКГС и другие данные последовательности в информации о презентации.
[00312] Когда данные для обучения 170 включают последовательности с различной длиной аминокислот, кодирующий модуль 314 может дополнительно кодировать пептиды в векторы равной длины путем добавления символа PAD для расширения заданного алфавита. Например, это может быть выполнено путем добавления слева остатков пептидных последовательностей с символом PAD до тех пор, пока длина пептидной последовательности не достигнет пептидной последовательности с наибольшей длиной в данных для обучения 170. Таким образом, когда пептидная последовательность с наибольшей длиной имеет аминокислоты kmax, кодирующий модуль 314 численно представляет каждую последовательность в виде вектора строк из (20+1)⋅kmax - элементов. В качестве примера, для расширенного алфавита {PAD, А, С, D, Е, F, G, Н, I, К, L, М, N, Р, Q, R, S, Т, V, W, Y } и максимальной длиной аминокислоты ктах=5, тот же пример пептидной последовательности EAF из 3 аминокислот может быть представлен вектором строк из 105 элементов pi=[1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0]. С-концевая фланкирующая последовательность ci или другие данные последовательности могут быть аналогично закодированы, как описано выше. Таким образом, каждая независимая переменная или столбец в пептидной последовательности pi или ci представляет присутствие определенной аминокислоты в определенном положении последовательности.
[00313] Хотя вышеупомянутый способ кодирования данных последовательности описан в отношении последовательностей, имеющих аминокислотные последовательности, способ можно аналогичным образом распространить на другие типы данных последовательности, такие как данные последовательности ДНК или РНК и тому подобное.
[00314] Кодирующий модуль 314 также кодирует один или более аллелей ГКГС ai для объекта данных i как вектор строки из m элементов, в котором каждому элементу h=1, 2, …, m соответствует уникальный идентифицированный аллель ГКГС. Элементы, соответствующие аллелям ГКГС, идентифицированным для объекта данных i, имеют значение 1. В противном случае остальные элементы имеют значение 0. В качестве примера, аллели HLA-B*07:02 и HLA-C* 01:03 для объекта данных i, соответствующие многоаллельной клеточной линии среди m=4 уникальных идентифицированных типов аллелей ГКГС {HLA-A*01:01, HLA-C*01: 08, HLA-B*07: 02, HLA-C*01: 03} могут быть представлены вектором строки из 4 элементов ai=[0 0 1 1], в которых 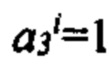 и
и 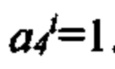 . Хотя пример описан в данном документе с 4 идентифицированными типами аллелей ГКГС, на практике количество типов аллелей ГКГС может быть сотни или тысячи. Как обсуждалось ранее, каждый объект данных i обычно содержит не более 6 различных типов аллелей ГКГС в связи с пептидной последовательностью pi.
. Хотя пример описан в данном документе с 4 идентифицированными типами аллелей ГКГС, на практике количество типов аллелей ГКГС может быть сотни или тысячи. Как обсуждалось ранее, каждый объект данных i обычно содержит не более 6 различных типов аллелей ГКГС в связи с пептидной последовательностью pi.
[00315] Кодирующий модуль 314 также кодирует метку _yi для каждого объекта данных i как двоичную переменную, имеющую значения из совокупности {0, 1}, в котором значение 1 указывает на то, что пептид xi был презентирован одним из ГКГС-ассоциированных аллелей ai, а значение 0 указывает на то, что пептид xi не был презентирован ни одним из ассоциированных аллелей ГКГС ai. Когда зависимая переменная yi представляет ионный ток масс-спектрометрии, кодирующий модуль 314 может дополнительно масштабировать значения с использованием различных функций, таких как логарифмическая функция, имеющая диапазон [-∞, ∞] для значений ионного тока между [0, ∞].
[00316] Кодирующий модуль 314 может представлять пару аллель-взаимодействующих переменных 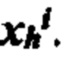 для пептида pi и ГКГС-ассоциированного аллеля h в качестве вектора строк, в котором числовые презентации аллель-взаимодействующих переменных конкатенируются один за другим. Например, кодирующий модуль 314 может представлять
для пептида pi и ГКГС-ассоциированного аллеля h в качестве вектора строк, в котором числовые презентации аллель-взаимодействующих переменных конкатенируются один за другим. Например, кодирующий модуль 314 может представлять 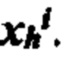 как вектор строки, равный [pi],
как вектор строки, равный [pi], 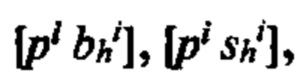 или
или 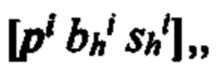 где
где 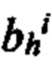 - прогноз аффинности связывания для пептида pi и ГКГС-ассоциированного аллеля h, и аналогично для
- прогноз аффинности связывания для пептида pi и ГКГС-ассоциированного аллеля h, и аналогично для 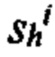 для стабильности. Альтернативно, одна или более комбинаций аллель-взаимодействующие переменные могут храниться отдельно (например, в виде отдельных векторов или матриц).
для стабильности. Альтернативно, одна или более комбинаций аллель-взаимодействующие переменные могут храниться отдельно (например, в виде отдельных векторов или матриц).
[00317] В одном случае кодирующий модуль 314 представляет информацию об аффинности связывания путем включения измеренных или прогнозируемых значений для аффинности связывания в аллель-взаимодействующие переменные 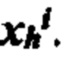
[00318] В одном случае кодирующий модуль 314 представляет информацию о стабильности связывания путем включения измеренных или прогнозируемых значений для стабильности связывания в аллель-взаимодействующие переменные 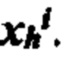
[00319] В одном случае кодирующий модуль 314 представляет информацию о скорости связывания путем включения измеренных или прогнозируемых значений для скорости связывания в аллель-взаимодействующие переменные 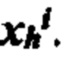 ,
,
[00320] В одном случае кодирующий модуль 314 представляет длину пептида в виде вектора 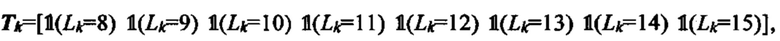 где
где  является индикаторной функцией, a Lk обозначает длину пептида pk. Вектор Tk может быть включен в аллель-взаимодействующие переменные
является индикаторной функцией, a Lk обозначает длину пептида pk. Вектор Tk может быть включен в аллель-взаимодействующие переменные 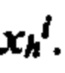
[00321] В одном случае кодирующая модуль 314 представляет информацию об экспрессии РНК аллелей ГКГС путем включения уровней экспрессии аллелей ГКГС на основе РНК-секвенирования в аллель-взаимодействующие переменные 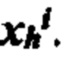
[00322] Аналогично, кодирующий модуль 314 может представлять аллель-невзаимодействующие переменные wi в качестве вектора строк, в котором числовые презентации аллель-невзаимодействующих переменных объединяются один за другим. Например, wi может быть вектором строки, равным [ci] или [ci mi wi], в котором wi представляет собой вектор строк, представляющий любые другие аллель-невзаимодействующие переменные в дополнение к С-концевой фланкирующей последовательности пептида pi и количественного измерения мРНК mi, связанного с пептидом. Альтернативно, одна или более комбинаций аллель-невзаимодействующих переменных могут храниться отдельно (например, в виде отдельных векторов или матриц).
[00323] В одном случае кодирующий модуль 314 представляет интенсивность обмена исходного белка для пептидной последовательности путем включения интенсивности обмена или периода полувыведения в аллель-невзаимодействующие переменные wi.
[00324] В одном случае кодирующий модуль 314 представляет длину исходного белка или изоформы путем включения длины белка в аллель-невзаимодействующие переменные wi.
[00325] В одном случае кодирующий модуль 314 представляет активацию иммунопротеасомы путем включения среднего значения экспрессии специфических к иммунопротеасоме субъединиц протеасомы, включая субъединицы β1i, β2i, β5i в аллель-невзаимодействующие переменные wi.
[00326] В одном случае кодирующий модуль 314 представляет собой количество РНК-секвенированного исходного белка пептида или гена или транскрипта пептида (количественно определяемого в единицах FPKM, ТРМ с помощью таких методов, как RSEM), может включать количества исходного белка в аллель-невзаимодействующие переменные wi.
[00327] В одном случае кодирующий модуль 314 представляет вероятность того, что транскрипт исходного пептида подвергнется нонсенс-опосредованному расщеплению (NMD), как оценивается моделью, например, в Rivas et. al. Science, 2015 путем включения этой вероятности в аллель-невзаимодействующие переменные wi.
[00328] В одном случае кодирующий модуль 314 представляет статус активации генного модуля или сигнального пути, оцененного посредством РНК-секвенирования, например, путем количественной оценки экспрессии генов в сигнальном пути в единицах ТРМ с использованием, например, RSEM для каждого из генов в сигнальном пути, затем вычисляет сводную статистику, например, среднее, через гены в пути. Среднее значение может быть включено в аллель-невзаимодействующие переменные wi.
[00329] В одном случае кодирующий модуль 314 представляет число копий исходного гена путем включения числа копий в аллель-невзаимодействующие переменные wi.
[00330] В одном случае кодирующий модуль 314 представляет аффинность связывания ТАР путем включения измеренной или спрогнозированной аффинности связывания ТАР (например, в наномолярных единицах) в аллель-невзаимодействующие переменные wi.
[00331] В одном случае кодирующий модуль 314 представляет уровни экспрессии ТАР путем включения уровней экспрессии ТАР, измеренных при помощи РНК-секвенирования (и количественно определенные в единицах ТРМ, например, RSEM), в аллель-невзаимодействующие переменные wi.
[00332] В одном случае кодирующий модуль 314 представляет опухолевые мутации как вектор индикаторных переменных (то есть dk=1, если пептид pk происходит из образца с мутацией KRAS G12D, и в противном случае равно 0) в аллель-невзаимодействующих переменных wi.
[00333] В одном случае кодирующий модуль 314 представляет полиморфизмы зародышевой линии в генах презентации антигенов в качестве вектора индикаторных переменных (то есть dk=1, если пептид pk происходит из образца со специфическим полиморфизмом зародышевой линии в ТАР). Эти индикаторные переменные могут быть включены в аллель-невзаимодействующие переменные wi.
[00334] В одном случае кодирующий модуль 314 представляет тип опухоли в виде одного из вектора прямого кодирования по кодовому набору типов опухоли (например, НМРЛ, меланома, колоректальный рак и т.д.). Эти переменные прямого кодирования могут быть включены в аллель-невзаимодействующие переменные wi.
[00335] В одном случае кодирующий модуль 314 представляет суффиксы аллелей ГКГС путем обработки 4-символьных аллелей HLA с различными суффиксами. Например, HLA-A*24:09N считается другим аллелем HLA-A*24:09 для целей модели. Альтернативно, вероятность презентации с помощью аллеля ГКГС с суффиксом N может быть установлена равной нулю для всех пептидов, поскольку аллели HLA, заканчивающиеся на суффикс N, не экспрессируются.
[00336] В одном случае кодирующий модуль 314 представляет собой подтип опухоли в виде одного из вектора прямого кодируемого по кодовому набору подтипов опухоли (например, аденокарцинома легкого, плоскоклеточная карцинома легкого и т.д.). Эти кодируемые прямо переменные могут быть включены в аллель-невзаимодействующие переменные wi.
[00337] В одном случае кодирующий модуль 314 представляет анамнез курения как двоичную индикаторную переменную (dk=1, если у пациента есть анамнез курения, и 0 в противном случае), который может быть включен в аллель-невзаимодействующие переменные wi. Альтернативно, анамнез курения может быть закодирован одной из переменной прямого кодирования по кодовому набору тяжести курения. Например, статус курения можно оценивать по шкале 1-5, где 1 обозначает некурящих, а 5 указывает на злостных курильщиков. Поскольку анамнез курения в первую очередь относится к опухолям легких, при обучении модели на нескольких типах опухолей эта переменная также может быть определена как равная 1, если пациент имеет анамнез курения, а тип опухоли - опухоли легких, а в противном случае - ноль.
[00338] В одном случае кодирующий модуль 314 представляет историю солнечных ожогов в виде двоичной индикаторной переменной (dk=1, если пациент имеет анамнез сильного солнечного ожога и 0 в противном случае), который может быть включен в аллель-невзаимодействующие переменные wi. Поскольку сильный солнечный ожог в первую очередь относится к меланомам, при обучении модели по множественным типам опухолей, эту переменную можно также определить равной 1, если у пациента есть тяжелый загар, а тип опухоли - меланома, а в противном случае - ноль.
[00339] В одном случае кодирующий модуль 314 представляет распределение уровней экспрессии конкретного гена или транскрипта для каждого гена или транскрипта в геноме человека в виде сводной статистики (например, средней, медианной) распределения уровней экспрессии с использованием справочных баз данных таких как TCGA. В частности, для пептида pk в образце с меланомой как типа опухоли мы можем включать не только измеренный уровень генной или транскрипционной экспрессии гена или транскрипта исходного пептид pk в аллель-невзаимодействующие переменные wi, но также среднее значение и/или медиана генной или транскрипционной экспрессии гена или транскрипта исходного пептида pk в меланомах, как измерено при помощи TCGA.
[00340] В одном случае кодирующий модуль 314 представляет собой тип мутации в качестве одной из переменной прямого кодирования по алфавиту типов мутаций (например, миссенс, сдвиг рамки считывания, индуцирование NMD и т.д.). Эти кодируемые прямо переменные могут быть включены в аллель-невзаимодействующие переменные wi.
[00341] В одном случае кодирующий модуль 314 представляет характеристики уровня белка в виде значения аннотации (например, длины 5 -НТО) исходного белка в аллель-невзаимодействующие переменные wi. В другом случае кодирующий модуль 314 кодирования представляет собой аннотации остаточного уровня исходного белка для пептида pk, включающие индикаторную переменную, которая равна 1, если пептид pk перекрывается с мотивом спирали и 0 в противном случае или равен 1, если пептид pk полностью содержится внутри спирального мотива в аллель-невзаимодействующие переменные wi. В другом случае признак, представляющий долю остатков в пептиде pk, которые содержатся в аннотации спирального мотива, может быть включен в аллель-невзаимодействующие переменные wi.
[00342] В одном случае кодирующий модуль 314 представляет тип белков или изоформ в протеоме человека в качестве индикаторного вектора ok, который имеет длину, равную количеству белков или изоформ в протеоме человека, и соответствующий элемент ok равен 1, если пептид pk происходит от белка i, и равен 0 в противном случае.
[00343] Кодирующий модуль 314 также может представлять общую совокупность переменных zi для пептида pi и ГКГС-ассоциированного аллеля h в качестве вектора строк, в котором числовые презентации аллель-взаимодействующих переменных xi, и аллель-невзаимодействующие переменные wi конкатенируются один за другим. Например, кодирующий модуль 314 может представлять 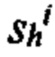 как вектор строки, равный [
как вектор строки, равный [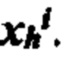 wi] или [wi
wi] или [wi 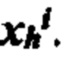 ].
].
VIII. Обучающий модуль
[00344] Обучающий модуль 316 создает одну или более моделей презентации, которые генерируют вероятности того, будут ли пептидные последовательности презентированы аллелями ГКГС, ассоциированными с пептидными последовательностями. В частности, с учетом пептидной последовательности pk и совокупности аллелей ГКГС ak, ассоциированных с пептидной последовательностью pk каждая модель презентации генерирует оценку uk, указывающую вероятность того, что пептидная последовательность pk будет презентирована одним или более связанными аллелями ГКГС ak.
VIII.A. Общие сведения
[00345] Обучающий модуль 316 создает еще одну модель презентации, основанную на совокупностях данных для обучения, сохраненной в хранилище 170, сгенерированной из информации презентации, сохраненной в 165. Как правило, независимо от конкретного типа модели презентации все модели презентации фиксируют зависимость между независимыми переменными и зависимыми переменными в данных для обучения 170, так что функция потерь минимизируется. В частности, функция потерь 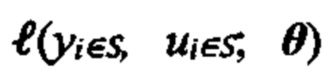 представляет несоответствия между значениями зависимых переменных yi∈s для одного или более объектов данных S в данных 170 обучения и предполагаемых вероятностей ui∈s для объектов данных S, сгенерированных при помощи модели презентации. В одном конкретном варианте осуществления, упомянутом в оставшейся части описания, функция потерь (yi∈s, ui∈s; θ) является отрицательной логарифмической функцией правдоподобия, заданной уравнением (1а), указанной ниже:
представляет несоответствия между значениями зависимых переменных yi∈s для одного или более объектов данных S в данных 170 обучения и предполагаемых вероятностей ui∈s для объектов данных S, сгенерированных при помощи модели презентации. В одном конкретном варианте осуществления, упомянутом в оставшейся части описания, функция потерь (yi∈s, ui∈s; θ) является отрицательной логарифмической функцией правдоподобия, заданной уравнением (1а), указанной ниже:
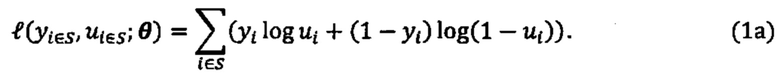
Однако на практике может использоваться другая функция потерь. Например, когда сделаны прогнозы для ионного тока масс-спектрометрии, функция потерь представляет собой среднеквадратичные потери, заданные уравнением 1b, как указано ниже:
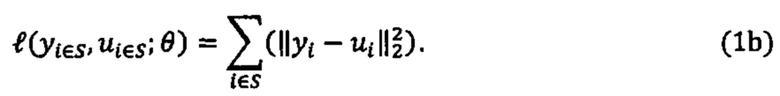
[00346] Модель презентации может быть параметрической моделью, в которой один или более параметров в математически θ определяют зависимость между независимыми переменными и зависимыми переменными. Как правило, различные параметры моделей презентации параметрического типа, которые минимизируют функцию потерь (yi∈s, ui∈s, θ), определяются с помощью методов численной оптимизации, основанных на градиенте, таких как пакетные градиентные методы, методы стохастического градиента, и тому подобное. Альтернативно, модель презентации может быть непараметрической моделью, в которой структура модели определяется из данных для обучения 170 и не является строго основанной на фиксированной совокупности параметров.
VIII.B. Модели для каждого аллеля
[00347] Обучающий модуль 316 может создавать модели презентации для прогнозирования вероятности презентации пептидов на основе каждого аллеля. В этом случае модуль для обучения 316 может обучать модели презентации на основе объектов данных S в данных для обучения 170, получаемых из клеток, экспрессирующих одиночные аллели ГКГС.
[00348] В одном варианте осуществления модуль для обучения 316 моделирует предполагаемую вероятность презентации uk для пептида pk для конкретного аллеля h путем:
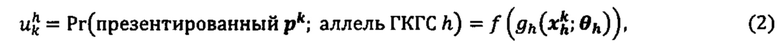
где пептидная последовательность 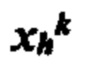 обозначает кодированные аллель-взаимодействующие переменные для пептида pk и соответствующий аллель ГКГС h, ƒ(⋅) представляет собой любую функцию и в данном документе всюду упоминается как функция преобразования для удобства описания. Кроме того, gh(⋅) представляет собой любую функцию, в данном документе всюду упоминается как функция зависимости для удобства описания и генерирует оценки зависимостей для аллель-взаимодействующих переменных
обозначает кодированные аллель-взаимодействующие переменные для пептида pk и соответствующий аллель ГКГС h, ƒ(⋅) представляет собой любую функцию и в данном документе всюду упоминается как функция преобразования для удобства описания. Кроме того, gh(⋅) представляет собой любую функцию, в данном документе всюду упоминается как функция зависимости для удобства описания и генерирует оценки зависимостей для аллель-взаимодействующих переменных 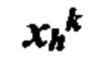 на основе совокупности параметров θh, определенных для аллеля h. Значения для совокупности параметров θh для каждого аллеля ГКГС h могут быть определены путем минимизации функции потерь по отношению к θh, где i представляет собой каждый объект в подсовокупности S данных для обучения 170, полученных из клеток, экспрессирующих один аллель ГКГС h.
на основе совокупности параметров θh, определенных для аллеля h. Значения для совокупности параметров θh для каждого аллеля ГКГС h могут быть определены путем минимизации функции потерь по отношению к θh, где i представляет собой каждый объект в подсовокупности S данных для обучения 170, полученных из клеток, экспрессирующих один аллель ГКГС h.
[00349] Результат функции зависимости  представляет собой оценку зависимости для аллеля ГКГС h, указывающую, будет ли аллель ГКГС h соответствовать соответствующему неоантигену на основе по меньшей мере аллель-взаимодействующих признаков
представляет собой оценку зависимости для аллеля ГКГС h, указывающую, будет ли аллель ГКГС h соответствовать соответствующему неоантигену на основе по меньшей мере аллель-взаимодействующих признаков 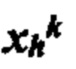 и, в частности, на основе положений аминокислот пептидной последовательности пептида pk. Например, оценка зависимости для аллеля ГКГС h может иметь высокое значение, если аллель ГКГС h, вероятно, будет представлять пептид pk и может иметь низкое значение, если презентация маловероятна. Функция преобразования ƒ(⋅) преобразует входные данные и, более конкретно, преобразует оценку зависимости, генерируемую
и, в частности, на основе положений аминокислот пептидной последовательности пептида pk. Например, оценка зависимости для аллеля ГКГС h может иметь высокое значение, если аллель ГКГС h, вероятно, будет представлять пептид pk и может иметь низкое значение, если презентация маловероятна. Функция преобразования ƒ(⋅) преобразует входные данные и, более конкретно, преобразует оценку зависимости, генерируемую 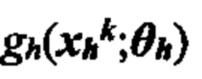 в этом случае, к соответствующему значению, чтобы указать вероятность того, что пептид pk будет презентирован аллелем ГКГС.
в этом случае, к соответствующему значению, чтобы указать вероятность того, что пептид pk будет презентирован аллелем ГКГС.
[00350] В одном конкретном варианте осуществления, упомянутом в оставшейся части описания, ƒ(⋅) является функцией, имеющей диапазон в пределах [0, 1] для соответствующего диапазона доменов. В одном примере ƒ(⋅) представляет собой логистическую функцию, заданную формулой:

В качестве другого примера, ƒ(⋅) также может быть функцией гиперболического тангенса, заданной формулой:

если значения для домена z равны или больше 0. Альтернативно, когда сделаны прогнозы для ионного тока масс-спектрометрии, которые имеют значения вне диапазона [0, 1], ƒ(⋅) может представлять собой любую функцию, такую как функция тождественности, экспоненциальная функция, лог-функция и т.п.
[00351] Таким образом, вероятность каждого аллеля того, что пептидная последовательность pk будет презентирована аллелем ГКГС h, может быть сгенерирована путем применения функции зависимости gh(⋅) для аллеля ГКГС h к кодированной версии пептидной последовательности pk для генерации соответствующей оценки зависимости. Показатель зависимости может быть преобразован при помощи функции преобразования ƒ(⋅) для генерации вероятности каждого аллеля так, что пептидная последовательность pk будет презентирована аллелем ГКГС h.
VIII.B.1 Функции зависимости для аллель-взаимодействующих переменных
[00352] В одной конкретной реализации, упомянутой в описании, функция зависимости gh(⋅) представляет собой линейную функцию, заданную формулой:

которая линейно объединяет каждую аллель-взаимодействующую переменную в 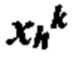 с соответствующим параметром в совокупности параметров θh, определенной для ГКГС-ассоциированного аллеля h.
с соответствующим параметром в совокупности параметров θh, определенной для ГКГС-ассоциированного аллеля h.
[00353] В другой конкретной реализации, упомянутой в описании, функция зависимости gh(⋅) представляет собой сетевую функцию, заданную формулой:
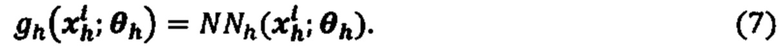
представленной сетевой моделью NNh(⋅), имеющей ряд узлов, расположенных в одном или более слоях. Узел может быть соединен с другими узлами через соединения, каждое из которых имеет связанный параметр в совокупности параметров θh. Значение на одном конкретном узле может быть представлено в виде суммы значений узлов, присоединенных к конкретному узлу, оцененных по соответствующему параметру, отображаемому при помощи активационной функции, связанной с конкретным узлом. В отличие от линейной функции, сетевые модели обладают преимуществом, поскольку модель презентации может включать нелинейность и обработку данных, имеющих разные длины аминокислотных последовательностей. В частности, посредством нелинейного моделирования сетевые модели могут охватывать взаимодействие между аминокислотами в разных положениях в пептидной последовательности и как это взаимодействие влияет на презентацию пептида.
[00354] В общем, сетевые модели NNh(⋅) могут быть структурированы как сети прямого распространения, такие как искусственные нейронные сети (ANN), сверточные нейронные сети (CNN), глубокие нейронные сети (DNN) и/или рекуррентные сети, такие как сети с долгой краткосрочной памятью (LSTM), двунаправленные рекуррентные сети, глубокие двунаправленные рекуррентные сети и тому подобное.
[00355] В одном случае, упомянутом в оставшейся части описания, каждый аллель ГКГС в h=1, 2, …, m связан с отдельной сетевой моделью, a NNh(⋅) обозначает выходные данные из сетевой модели, связанной с аллелью ГКГС h.
[00356] На фиг. 5 изображена иллюстративная сетевая модель NN3(⋅) в ассоциации с произвольным аллелем ГКГС h=3. Как изображено на фиг. 5, сетевая модель NN3(⋅) для аллеля ГКГС h=3 содержит три входных узла в слое  четыре узла в слое
четыре узла в слое  , два узла в слое
, два узла в слое 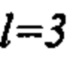 и один выходной узел в слое
и один выходной узел в слое 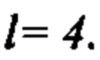 Сетевая модель NN3(⋅) связана с совокупностью из десяти параметров θ3(1), θ3(2), …, θ3(10). Сетевая модель NN3(⋅) принимает входные значения (отдельные объекты данных, включая закодированные данные полипептидной последовательности и любые другие данные для обучения) для трех аллель-взаимодействующих переменных
Сетевая модель NN3(⋅) связана с совокупностью из десяти параметров θ3(1), θ3(2), …, θ3(10). Сетевая модель NN3(⋅) принимает входные значения (отдельные объекты данных, включая закодированные данные полипептидной последовательности и любые другие данные для обучения) для трех аллель-взаимодействующих переменных 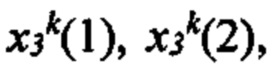 и
и 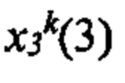 для аллеля ГКГС h=3 и выводит значение
для аллеля ГКГС h=3 и выводит значение 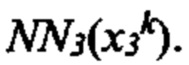
[00357] В другом случае идентифицированные аллели ГКГС h=l, 2, …, m связаны с моделью с единой сетью NNH(⋅), и NNh(⋅) обозначает одно или более выходных данных модели с единой сетью, связанной с аллелем ГКГС h. В таком случае совокупность параметров θh может соответствовать совокупности параметров для модели с единой сетью, и, таким образом, совокупность параметров θh может быть совместно использована всеми аллелями ГКГС.
[00358] На фиг. 6А изображена иллюстративная сетевая модель NNH(⋅), совместно используемая аллелями ГКГС h=l, 2, …, m. Как изображено на фиг. 6А, сетевая модель NNH(⋅) содержит т выходных узлов, каждый из которых соответствует аллелю ГКГС. Сетевая модель NN3(⋅) принимает аллель-взаимодействующие переменные  для аллеля ГКГС h=3 и выводит значения m, включая значение
для аллеля ГКГС h=3 и выводит значения m, включая значение 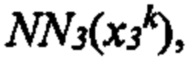 соответствующее аллелю ГКГС h=3.
соответствующее аллелю ГКГС h=3.
[00359] В еще одном случае модель с единой сетью NNH(⋅) может представлять собой сетевую модель, которая выводит оценку зависимости, учитывая аллель-взаимодействующие переменные  и кодированную белковую последовательность dh аллеля ГКГС h. В таком случае совокупность параметров θh может снова соответствовать совокупности параметров для модели с единой сетью, и, таким образом, совокупность параметров θh может быть совместно использована всеми аллелями ГКГС. Таким образом, в таком случае NNh(⋅) может обозначать выходные данные модели с единой сетью NNH(⋅) с учетом входных данных
и кодированную белковую последовательность dh аллеля ГКГС h. В таком случае совокупность параметров θh может снова соответствовать совокупности параметров для модели с единой сетью, и, таким образом, совокупность параметров θh может быть совместно использована всеми аллелями ГКГС. Таким образом, в таком случае NNh(⋅) может обозначать выходные данные модели с единой сетью NNH(⋅) с учетом входных данных 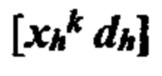 в модель с единой сетью. Такая сетевая модель является предпочтительной, поскольку вероятности презентации пептидов для аллелей ГКГС, которые были неизвестны в данных для обучения, могут быть спрогнозированы только путем идентификации их белковой последовательности.
в модель с единой сетью. Такая сетевая модель является предпочтительной, поскольку вероятности презентации пептидов для аллелей ГКГС, которые были неизвестны в данных для обучения, могут быть спрогнозированы только путем идентификации их белковой последовательности.
[00360] На фиг. 6В изображена иллюстративная сетевая модель NNH(⋅), совместно используемая аллелями ГКГС. Как изображено на фиг. 6В, сетевая модель NNH(⋅) принимает аллель-взаимодействующие переменные и белковую последовательность аллеля ГКГС h=3 в качестве входных данных, и выводит оценку зависимости 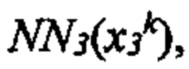 соответствующую аллелю ГКГС h=3.
соответствующую аллелю ГКГС h=3.
[00361] В еще одном случае функция зависимости gh(⋅) может быть выражена как:
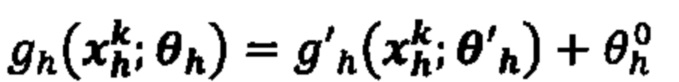
где  представляет собой линейную функцию с совокупностью параметров
представляет собой линейную функцию с совокупностью параметров  , сетевой функции или тому подобное, с параметром смещения
, сетевой функции или тому подобное, с параметром смещения  в совокупности параметров для аллель-взаимодействующих переменных для аллеля ГКГС, что представляет собой исходную вероятность презентации аллелем ГКГС h.
в совокупности параметров для аллель-взаимодействующих переменных для аллеля ГКГС, что представляет собой исходную вероятность презентации аллелем ГКГС h.
[00362] В другой реализации параметр смещения  может быть совместно использован в соответствии с семейством генов аллеля ГКГС h. То есть параметр смещения
может быть совместно использован в соответствии с семейством генов аллеля ГКГС h. То есть параметр смещения 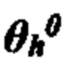 для аллеля ГКГС h может быть равен
для аллеля ГКГС h может быть равен 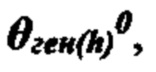 где ген (h) принадлежит семейству генов аллеля ГКГС h. Например, аллели ГКГС HLA-A*02:01, HLA-A*02:02, и HLA-A*02:03 могут быть отнесены к семейству генов «HLA-А», а параметр смещения
где ген (h) принадлежит семейству генов аллеля ГКГС h. Например, аллели ГКГС HLA-A*02:01, HLA-A*02:02, и HLA-A*02:03 могут быть отнесены к семейству генов «HLA-А», а параметр смещения 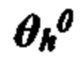 для каждого из этих аллелей ГКГС может быть совместно использован.
для каждого из этих аллелей ГКГС может быть совместно использован.
[00363] Возвращаясь к уравнению (2), в качестве примера, вероятность того, что пептид pk будет презентирован аллелем ГКГС h=5, среди m=4 различных идентифицированных аллелей ГКГС с использованием линейной функции зависимости gh(⋅) может быть получена путем:
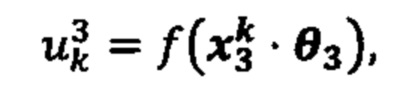
где 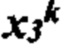 представляют собой идентифицированные аллель-взаимодействующие переменные для аллеля ГКГС h=3, а θ3 представляют собой совокупность параметров, определенных для аллеля ГКГС h=3 с помощью минимизации функции потерь.
представляют собой идентифицированные аллель-взаимодействующие переменные для аллеля ГКГС h=3, а θ3 представляют собой совокупность параметров, определенных для аллеля ГКГС h=3 с помощью минимизации функции потерь.
[00364] В качестве другого примера, вероятность того, что пептид pk будет презентирован аллелем ГКГС h=3, среди m=4 различных идентифицированных аллелей ГКГС с использованием отдельных сетевых функций преобразования gh(⋅), может быть получена путем:
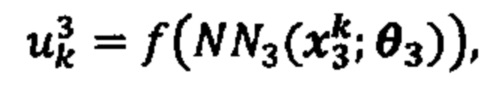
где 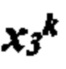 представляют собой идентифицированные аллель-взаимодействующие переменные для аллеля ГКГС h=3, и θ3 представляют собой совокупность параметров, определенных для сетевой модели NN3(⋅), связанной с аллелем ГКГС h=3.
представляют собой идентифицированные аллель-взаимодействующие переменные для аллеля ГКГС h=3, и θ3 представляют собой совокупность параметров, определенных для сетевой модели NN3(⋅), связанной с аллелем ГКГС h=3.
[00365] На фиг. 7 изображено генерирование вероятности презентации для пептида pk в ассоциации с аллелем ГКГС h=3 с использованием иллюстративной сетевой модели NN3(⋅). Как изображено на фиг. 7, сетевая модель NN3(⋅) принимает аллель-взаимодействующие переменные 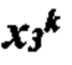 для аллеля ГКГС h=3 и генерирует выходные данные
для аллеля ГКГС h=3 и генерирует выходные данные 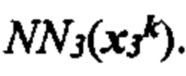 Результат отображается функцией ƒ(⋅) для генерации предполагаемой вероятности презентации uk.
Результат отображается функцией ƒ(⋅) для генерации предполагаемой вероятности презентации uk.
VIII.B.2. Аллель-невзаимодействующие переменные на аллель
[00366] В одной реализации модуль для обучения 316 включает аллель-невзаимодействующие переменные и модели предполагаемой вероятности презентации uk для пептида pk посредством:
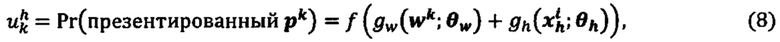
где wk обозначает кодированные аллель-невзаимодействующие переменные для пептида pk, gw(⋅) представляет собой функцию для аллель-невзаимодействующих переменных wk на основе совокупности параметров θw, определенных для аллель-невзаимодействующих переменных. В частности, значения для совокупности параметров θh для каждого аллеля ГКГС h и совокупности параметров θw для аллель-невзаимодействующих переменных можно определить, минимизируя функцию потерь по θh и θw„ где i - каждый объект в подсовокупности S данных для обучения 170, получаемых из клеток, экспрессирующих одиночные аллели ГКГС.
[00367] Результат функции зависимости gw(wk; θw) представляет собой оценку зависимости для аллель-невзаимодействующих переменных, указывающих, будет ли пептид pk презентирован одним или более аллелями ГКГС на основе воздействия аллель-невзаимодействующих переменных. Например, оценка зависимости для аллель-невзаимодействующих переменных может иметь высокое значение, если пептид pk связан с С-концевой фланкирующей последовательностью, которая, как известно, положительно влияет на презентацию пептида pk, и может иметь низкое значение, если пептид pk связан с С-концевой фланкирующей последовательностью, которая, как известно, отрицательно влияет на презентацию пептида pk.
[00368] Согласно уравнению (8), вероятность каждого аллеля того, что пептидная последовательность pk будет презентирована аллелем ГКГС h, может быть сгенерирована путем применения функции gh(⋅) для аллеля ГКГС h к кодированной версии пептидной последовательности pk для генерации соответствующей оценки зависимости для аллель-взаимодействующих переменных. Функция gw(⋅) для аллель-невзаимодействующих переменных также применяется к кодированной версии аллель-невзаимодействующих переменных для генерации оценки зависимости для аллель-невзаимодействующих переменных. Оба показателя объединяются, и комбинированный результат преобразуется функцией преобразования ƒ(⋅) для генерации вероятности на аллель так, что пептидная последовательность pk будет презентирована аллелем ГКГС h.
[00369] В качестве альтернативы, модуль для обучения 316 может включать аллель-невзаимодействующие переменные wk в прогнозировании путем добавления аллель-невзаимодействующих переменных wk к аллель-взаимодействующим переменным 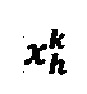 в уравнении (2). Таким образом, вероятность презентации может быть задана формулой:
в уравнении (2). Таким образом, вероятность презентации может быть задана формулой:
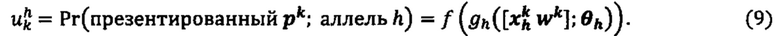
VIII.B.3 Функции зависимости для аллель-невзаимодействующих переменных
[00370] Аналогично функции зависимости gh(⋅) для аллель-взаимодействующих переменных, функция зависимости gw(⋅) для аллель-невзаимодействующих переменных может быть линейной функцией или сетевой функцией, в которой отдельная сетевая модель связана с аллель-невзаимодействующими переменными wk.
[00371] В частности, функция зависимости gw(⋅) представляет собой линейную функцию, заданную формулой:
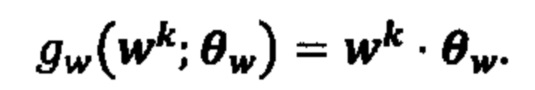
которая линейно объединяет аллель-невзаимодействующие переменные в wk с соответствующим параметром в совокупности параметров θw.
[00372] Функция зависимости gw(⋅) также может представлять собой сетевую функцию, заданную формулой:
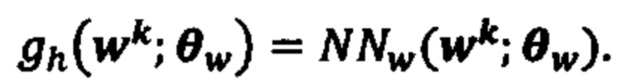
представленной сетевой моделью NNw(⋅), имеющей связанный параметр в совокупности параметров θw..
[00373] В другом случае функция зависимости gw(⋅)для аллель-невзаимодействующих переменных может быть задана формулой:
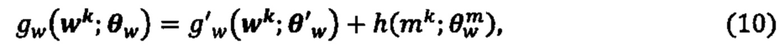
где 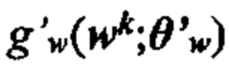 представляет собой линейную функцию, сетевую функцию с множеством аллель-невзаимодействующих параметров
представляет собой линейную функцию, сетевую функцию с множеством аллель-невзаимодействующих параметров 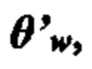 или тому подобное, mk представляет собой количественное измерение мРНК для пептида pk, h(⋅) представляет собой функцию, преобразующую количественное измерение, и
или тому подобное, mk представляет собой количественное измерение мРНК для пептида pk, h(⋅) представляет собой функцию, преобразующую количественное измерение, и 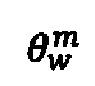 представляет собой параметр в совокупности параметров для аллель-невзаимодействующих переменных, которые объединены с количественным измерением мРНК для генерации оценки зависимости для количественного измерения мРНК. В одном конкретном варианте осуществления, упомянутом в оставшейся части описания, h(⋅) представляет собой лог-функцию, однако на практике h(⋅) может быть любой из множества различных функций.
представляет собой параметр в совокупности параметров для аллель-невзаимодействующих переменных, которые объединены с количественным измерением мРНК для генерации оценки зависимости для количественного измерения мРНК. В одном конкретном варианте осуществления, упомянутом в оставшейся части описания, h(⋅) представляет собой лог-функцию, однако на практике h(⋅) может быть любой из множества различных функций.
[00374] В еще одном случае функция зависимости gw(⋅) для аллель-невзаимодействующих переменных может быть задана формулой:
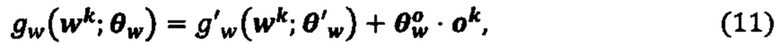
где 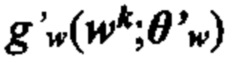 представляет собой линейную функцию, сетевую функцию с совокупностью аллель-невзаимодействующих параметров
представляет собой линейную функцию, сетевую функцию с совокупностью аллель-невзаимодействующих параметров 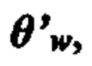 или тому подобное, ok представляет собой индикаторный вектор, описанный выше, представляющий белки и изоформы в протеоме человека для пептид pk, и
или тому подобное, ok представляет собой индикаторный вектор, описанный выше, представляющий белки и изоформы в протеоме человека для пептид pk, и 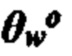 представляет собой совокупность параметров в совокупности параметров для аллель-невзаимодействующих переменных, которые комбинируются с индикаторным вектором. В одном варианте, когда размерность ok и совокупность параметров
представляет собой совокупность параметров в совокупности параметров для аллель-невзаимодействующих переменных, которые комбинируются с индикаторным вектором. В одном варианте, когда размерность ok и совокупность параметров 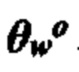 значительно высоки, член параметра регуляризации, такой как
значительно высоки, член параметра регуляризации, такой как 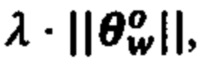 где ||⋅|| представляет собой норму L1, норму L2, комбинацию и т.п., можно добавить к функции потерь при определении значения параметров. Оптимальное значение гиперпараметра λ, можно определить с помощью соответствующих методов.
где ||⋅|| представляет собой норму L1, норму L2, комбинацию и т.п., можно добавить к функции потерь при определении значения параметров. Оптимальное значение гиперпараметра λ, можно определить с помощью соответствующих методов.
[00375] Возвращаясь к уравнению (8), в качестве примера, вероятность того, что пептид pk будет презентирован аллелем ГКГС h=3, среди m=4 различных идентифицированных аллелей ГКГС с использованием линейных функций преобразования gh(⋅), gw(⋅), может быть получена путем:

где wk представляют собой идентифицированные аллель-невзаимодействующие переменные для пептида pk, и θw представляют собой совокупность параметров, определенных для аллель-невзаимодействующих переменных.
[00376] В качестве другого примера, вероятность того, что пептид будет презентирован аллелем ГКГС h=3, среди m=4 различных идентифицированных аллелей ГКГС с использованием сетевых функций преобразования gh(⋅), gw(⋅), может быть получена путем:
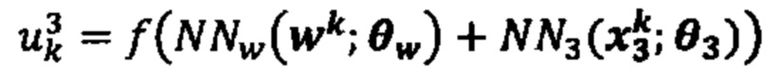
где wk представляют собой идентифицированные аллель-невзаимодействующие переменные для пептида pk, и θw представляют собой совокупность параметров, определенных для аллель-невзаимодействующих переменных.
[00377] На фиг. 8 изображено генерирование вероятности презентации для пептида pk в ассоциации с аллелем ГКГС h=3 с использованием иллюстративных сетевых моделей NN3(⋅) и NNw(⋅). Как изображено на фиг. 8, сетевая модель NN3(⋅) принимает аллель-взаимодействующие переменные 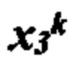 для аллеля ГКГС h=3 и генерирует выходные данные
для аллеля ГКГС h=3 и генерирует выходные данные 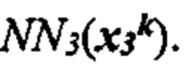 Сетевая модель NNw(⋅) принимает аллель-невзаимодействующие переменные wk для пептида pk и генерирует выходные данные NNw(wk). Результаты комбинируются и отображаются при помощи функции ƒ(⋅) для генерации предполагаемой вероятности презентации uk.
Сетевая модель NNw(⋅) принимает аллель-невзаимодействующие переменные wk для пептида pk и генерирует выходные данные NNw(wk). Результаты комбинируются и отображаются при помощи функции ƒ(⋅) для генерации предполагаемой вероятности презентации uk.
VIII.C. Многоаллельные модели
[00378] Модуль для обучения 316 также может создавать модели презентации для прогнозирования вероятностей презентации пептидов при многоаллельных параметрах, где присутствуют два или более аллелей ГКГС. В этом случае модуль для обучения 316 может обучать модели презентации на основе объектов данных S в данных для обучения 170, получаемых из клеток, экспрессирующих одиночные аллели ГКГС, из клеток, экспрессирующих множественные аллели ГКГС, или их комбинации.
VTH.C.1. Пример 1: Максимум моделей для каждого аллеля
[00379] В одном варианте осуществления модуль для обучения 316 моделирует предполагаемую вероятность презентации uk для пептида pk в ассоциации с совокупностью множественных аллелей Н в виде функции от вероятности презентации 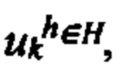 определенной для каждого из аллелей ГКГС h в совокупности Н, определяемой на основе клеток, экспрессирующих одиночные аллели, как описано выше в сочетании с уравнениями (2)-(11). В частности, вероятность презентации uk может представлять собой любую функцию
определенной для каждого из аллелей ГКГС h в совокупности Н, определяемой на основе клеток, экспрессирующих одиночные аллели, как описано выше в сочетании с уравнениями (2)-(11). В частности, вероятность презентации uk может представлять собой любую функцию  В одном варианте осуществления, как показано в уравнении (12), функция представляет собой функцию максимума, а вероятность презентации uk может быть определена как максимум вероятности презентации для каждого аллеля ГКГС h в совокупности Н.
В одном варианте осуществления, как показано в уравнении (12), функция представляет собой функцию максимума, а вероятность презентации uk может быть определена как максимум вероятности презентации для каждого аллеля ГКГС h в совокупности Н.
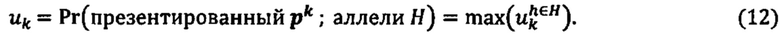
VIII.C.2. Пример 2.1; Модели функции сумм
[00380] В одном варианте осуществления модуль для обучения 316 моделирует предполагаемую вероятность презентации uk для пептида pk:
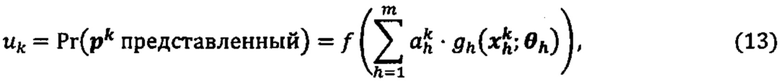
где элементы  равны 1 для множественных аллелей ГКГС Н, связанных с пептидной последовательностью pk и
равны 1 для множественных аллелей ГКГС Н, связанных с пептидной последовательностью pk и 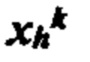 обозначают кодированные аллель-взаимодействующие переменные для пептида pk и соответствующие аллели ГКГС. Значения для совокупности параметров θh для каждого аллеля ГКГС h могут быть определены путем минимизации функции потерь по отношению к θh, где i представляет собой каждый объект в подсовокупности S данных для обучения 170, полученных из клеток, экспрессирующих одиночную аллель ГКГС, и/или клеток, экспрессирующих множество аллелей ГКГС. Функция зависимости gh может быть в форме любой из функций зависимости gh, введенных выше в разделах VIII.B.1.
обозначают кодированные аллель-взаимодействующие переменные для пептида pk и соответствующие аллели ГКГС. Значения для совокупности параметров θh для каждого аллеля ГКГС h могут быть определены путем минимизации функции потерь по отношению к θh, где i представляет собой каждый объект в подсовокупности S данных для обучения 170, полученных из клеток, экспрессирующих одиночную аллель ГКГС, и/или клеток, экспрессирующих множество аллелей ГКГС. Функция зависимости gh может быть в форме любой из функций зависимости gh, введенных выше в разделах VIII.B.1.
[00381] Согласно уравнению (13) вероятность презентации того, что пептидная последовательность pk будет презентирована одним или более аллелями ГКГС h, может быть сгенерирована путем применения функции зависимости gh(⋅) к кодированной версии пептидной последовательности pk для каждого из аллелей ГКГС Н для генерации соответствующей оценки для аллель-взаимодействующих переменных. Показатели для каждого аллеля ГКГС h комбинируются и преобразуются при помощи функции преобразования ƒ(⋅) для генерации вероятности презентации так, что последовательность пептида pk будет презентирована совокупностью аллелей ГКГС Н.
[00382] Модель презентации уравнения (13) отличается от модели на аллель уравнения (2) тем, что количество ассоциированных аллелей для каждого пептида pk может быть больше 1. Другими словами, более чем один элемент в 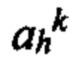 может иметь значения 1 для множественных аллелей ГКГС Н, связанных с пептидной последовательностью pk.
может иметь значения 1 для множественных аллелей ГКГС Н, связанных с пептидной последовательностью pk.
[00383] В качестве примера, вероятность того, что пептид pk будет презентирован аллелями ГКГС h=2, h=3, среди m=4 различных идентифицированных аллелей ГКГС с использованием линейных функций преобразования gh(⋅), может быть получена путем:
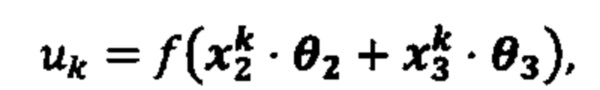
где 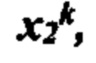
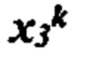 представляют собой идентифицированные аллель-взаимодействующие переменные для аллелей ГКГС h=2, h=3 и θ2, θ3 представляет собой совокупность параметров, определенных для аллелей ГКГС h=2, h=3.
представляют собой идентифицированные аллель-взаимодействующие переменные для аллелей ГКГС h=2, h=3 и θ2, θ3 представляет собой совокупность параметров, определенных для аллелей ГКГС h=2, h=3.
[00384] В качестве другого примера, вероятность того, что пептид pk будет презентирован аллелями ГКГС h=2, h=3, среди m=4 различных идентифицированных аллелей ГКГС с использованием сетевых функций преобразования gh(⋅), gw(⋅), может быть получена путем:
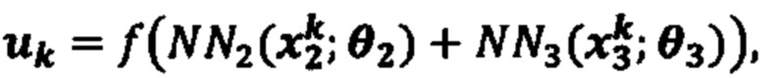
где NN2(⋅), NN3(⋅) представляют собой идентифицированные сетевые модели для аллелей ГКГС h=2, h=3 и θ2, θ3 представляют собой совокупность параметров, определяемых для аллелей ГКГС h=2, h=3.
[00385] На фиг. 9 изображено генерирование вероятности презентации для пептида pk в ассоциации с аллелями ГКГС h=2, h=3 с использованием иллюстративных сетевых моделей NN2(⋅) и NN3(⋅). Как изображено на фиг. 9, сетевая модель NN2(⋅) принимает аллель-взаимодействующие переменные 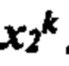 для аллеля ГКГС h=2 и генерирует выходные данные
для аллеля ГКГС h=2 и генерирует выходные данные 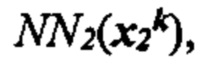 а сетевая модель NN3(⋅) принимает аллель-взаимодействующие переменные
а сетевая модель NN3(⋅) принимает аллель-взаимодействующие переменные 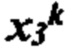 для аллеля ГКГС h=3 и генерирует выходные данные
для аллеля ГКГС h=3 и генерирует выходные данные 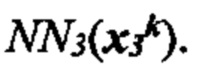 Результаты комбинируются и отображаются при помощи функции ƒ(⋅) для генерации предполагаемой вероятности презентации uk.
Результаты комбинируются и отображаются при помощи функции ƒ(⋅) для генерации предполагаемой вероятности презентации uk.
УШ.С.3. Пример 2.2: Модели функции сумм с аллель-невзаимодействующими переменными
[00386] В одной реализации модуль для обучения 316 включает аллель-невзаимодействующие переменные и модели предполагаемой вероятности презентации uk для пептида pk посредством:
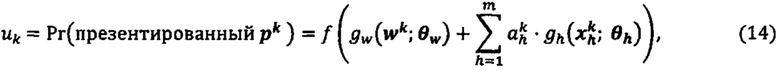
где wk обозначает кодированные аллель-невзаимодействующие переменные для пептида pk. В частности, значения для совокупности параметров θh для каждого аллеля ГКГС h и совокупности параметров θw для аллель-невзаимодействующих переменных можно определить, минимизируя функцию потерь по θh и θw„ где i - каждый объект в подсовокупности S данных для обучения 170, получаемых из клеток, экспрессирующих одиночные аллели ГКГС, и/или клеток, экспрессирующих множественные аллели ГКГС Функция зависимости gw может быть в форме любой из функций зависимости gw, введенных выше в разделах VIII.B.3.
[00387] Таким образом, согласно уравнению (14) вероятность презентации того, что пептидная последовательность pk будет презентирована одним или более аллелями ГКГС Н, может быть сгенерирована путем применения функции gh(⋅) к кодированной версии пептидной последовательности pk для каждого из аллелей ГКГС Н для генерации соответствующей оценки зависимости для аллель-взаимодействующих переменных для каждой аллели ГКГС h. Функция gw(⋅) для аллель-невзаимодействующих переменных также применяется к кодированной версии аллель-невзаимодействующих переменных для генерации оценки зависимости для аллель-невзаимодействующих переменных. Показатели объединяются, и комбинированный показатель преобразуется при помощи функции преобразования ƒ(⋅) для генерации вероятности презентации так, что пептидная последовательность pk будет презентирована аллелями ГКГС h.
[00388] В модели презентации уравнения (14) число ассоциированных аллелей для каждого пептида pk может быть больше 1. Другими словами, более чем один элемент в 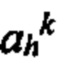 может иметь значения 1 для множественных аллелей ГКГС Н, связанных с пептидной последовательностью pk.
может иметь значения 1 для множественных аллелей ГКГС Н, связанных с пептидной последовательностью pk.
[00389] В качестве примера, вероятность того, что пептид pk будет презентирован аллелями ГКГС h=2, h=3, среди m=4 различных идентифицированных аллелей ГКГС с использованием линейных функций преобразования gh(⋅), gw(⋅) может быть получена путем:
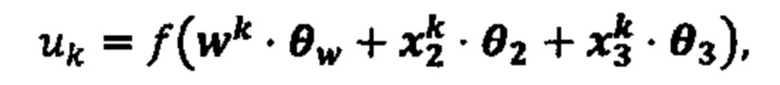
где wk представляют собой идентифицированные аллель-невзаимодействующие переменные для пептида pk, и θw представляют собой совокупность параметров, определенных для аллель-невзаимодействующих переменных.
[00390] В качестве другого примера, вероятность того, что пептид pk будет презентирован аллелями ГКГС h=2, h=3, среди m=4 различных идентифицированных аллелей ГКГС с использованием сетевых функций преобразования gh(⋅), gw(⋅), может быть получена путем:
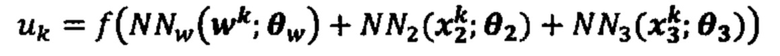
где wk представляют собой идентифицированные аллель-невзаимодействующие переменные для пептида pk, и θw представляют собой совокупность параметров, определенных для аллель-невзаимодействующих переменных.
[00391] На фиг. 10 изображено генерирование вероятности презентации для пептида pk в ассоциации с аллелями ГКГС h=2, h=3 с использованием иллюстративных сетевых моделей NN2(⋅), NN3(⋅), и NNw(⋅). Как изображено на фиг. 10, сетевая модель NN2(⋅) принимает аллель-взаимодействующие переменные 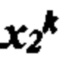 для аллеля ГКГС h=2 и генерирует выходные данные
для аллеля ГКГС h=2 и генерирует выходные данные 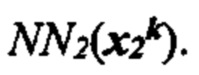 Сетевая модель NN3(⋅) принимает аллель-взаимодействующие переменные
Сетевая модель NN3(⋅) принимает аллель-взаимодействующие переменные  для аллеля ГКГС h=3 и генерирует выходные данные
для аллеля ГКГС h=3 и генерирует выходные данные 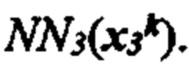 Сетевая модель NNw(⋅) принимает аллель-невзаимодействующие переменные wk для пептида pk и генерирует выходные данные NNw(wk). Результаты комбинируются и отображаются при помощи функции ƒ(⋅) для генерации предполагаемой вероятности презентации uk.
Сетевая модель NNw(⋅) принимает аллель-невзаимодействующие переменные wk для пептида pk и генерирует выходные данные NNw(wk). Результаты комбинируются и отображаются при помощи функции ƒ(⋅) для генерации предполагаемой вероятности презентации uk.
[00392] В качестве альтернативы, модуль для обучения 316 может включать аллель-невзаимодействующие переменные wk в прогнозировании путем добавления аллель-невзаимодействующих переменных wk аллель-взаимодействующим переменным 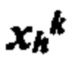 в уравнении (15). Таким образом, вероятность презентации может быть задана формулой:
в уравнении (15). Таким образом, вероятность презентации может быть задана формулой:
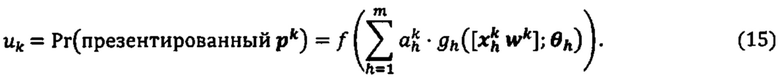
VIII.C.4. Пример 3.1: Модели с использованием неявных вероятностей для каждого аллеля
[00393] В другом варианте осуществления модуль для обучения 316 моделирует предполагаемую вероятность презентации uk для пептида pk:
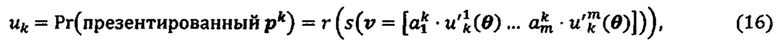
где элементы 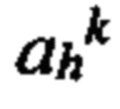 равны 1 для множественных аллелей ГКГС h∈Н, связанных с пептидной последовательностью pk,
равны 1 для множественных аллелей ГКГС h∈Н, связанных с пептидной последовательностью pk, 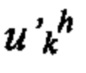 является неявной вероятностью презентации на аллель для аллеля ГКГС h, вектор ν представляет собой вектор, в котором элементу νh соответствует
является неявной вероятностью презентации на аллель для аллеля ГКГС h, вектор ν представляет собой вектор, в котором элементу νh соответствует 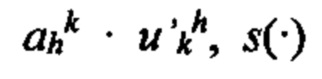 представляет собой функциональное преобразование элементов ν, и r(⋅)) представляет собой функция клиппирования, которая обрезает значение входных данных в заданный диапазон. Как описано ниже более подробно, s(⋅) может представлять собой функцию сложения или функцию второго порядка, но понятно, что в других вариантах осуществления s(⋅) может быть любой функцией, такой как функция максимума. Значения для совокупности параметров θ для неявных вероятностей на аллель могут быть определены путем минимизации функции потерь по θ, где i представляет собой каждый объект в подсовокупности 5 данных для обучения 170, генерируемых из клеток, экспрессирующих одиночные аллели ГКГС, и/или клеток, экспрессирующих множественные аллели ГКГС.
представляет собой функциональное преобразование элементов ν, и r(⋅)) представляет собой функция клиппирования, которая обрезает значение входных данных в заданный диапазон. Как описано ниже более подробно, s(⋅) может представлять собой функцию сложения или функцию второго порядка, но понятно, что в других вариантах осуществления s(⋅) может быть любой функцией, такой как функция максимума. Значения для совокупности параметров θ для неявных вероятностей на аллель могут быть определены путем минимизации функции потерь по θ, где i представляет собой каждый объект в подсовокупности 5 данных для обучения 170, генерируемых из клеток, экспрессирующих одиночные аллели ГКГС, и/или клеток, экспрессирующих множественные аллели ГКГС.
[00394] Вероятность презентации в модели презентации уравнения (17) моделируется как функция неявных вероятностей презентации для каждого аллеля 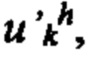 каждая из которых соответствует вероятности того, будет ли пептид pk презентирован отдельным аллелем ГКГС h. Неявная вероятность на аллель отличается от вероятности презентации на аллель в разделе VIII.B тем, что параметры для неявных вероятностей на аллель могут быть изучены из множества параметров аллели, в которых прямая связь между презентированным пептидом и соответствующим аллелем ГКСГ неизвестна, в дополнение к одноаллельным параметрам. Таким образом, при многоаллельных параметрах модель презентации может оценить не только, будет ли пептид pk презентирован совокупностью аллелей ГКГС H в целом, но также может обеспечивать отдельные вероятности
каждая из которых соответствует вероятности того, будет ли пептид pk презентирован отдельным аллелем ГКГС h. Неявная вероятность на аллель отличается от вероятности презентации на аллель в разделе VIII.B тем, что параметры для неявных вероятностей на аллель могут быть изучены из множества параметров аллели, в которых прямая связь между презентированным пептидом и соответствующим аллелем ГКСГ неизвестна, в дополнение к одноаллельным параметрам. Таким образом, при многоаллельных параметрах модель презентации может оценить не только, будет ли пептид pk презентирован совокупностью аллелей ГКГС H в целом, но также может обеспечивать отдельные вероятности 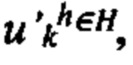 которые указывают, какой аллель ГКГС h наиболее вероятно презентирует пептид pk. Преимущество этого заключается в том, что модель презентации может генерировать неявные вероятности без данных обучения для клеток, экспрессирующих одиночные аллели ГКГС.
которые указывают, какой аллель ГКГС h наиболее вероятно презентирует пептид pk. Преимущество этого заключается в том, что модель презентации может генерировать неявные вероятности без данных обучения для клеток, экспрессирующих одиночные аллели ГКГС.
[00395] В одном конкретном варианте осуществления, упомянутом в оставшейся части описания, r(⋅) является функцией, имеющей диапазон [0, 1]. Например, r(⋅) может представлять собой функцию клиппирования:
r(z)=min(max(z, 0), 1),
где минимальное значение между z и 1 выбирается как вероятность презентации uk. В другом варианте осуществления r(⋅) представляет собой функцию гиперболического тангенса, заданную формулой:
r(z)=tanh(z)
если значения для домена z равны или больше 0.
VIII.C.5. Пример 3.2: Модель суммы функций
[00396] В одном конкретном варианте осуществления s(⋅) представляет собой функцию сложения, а вероятность презентации задается сложением неявных вероятностей презентации для каждого аллеля:
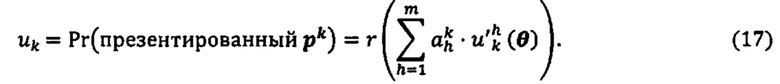
[00397] В одном варианте осуществления неявная вероятность презентации на аллель для аллеля ГКГС h генерируется посредством:

так что вероятность презентации оценивается по:

[00398] Согласно уравнению (19) вероятность презентации того, что пептидная последовательность pk будет презентирована одним или более аллелями ГКГС Н, может быть сгенерирована путем применения функции gh(⋅) к кодированной версии пептидной последовательности pk для каждого из аллелей ГКГС Н для генерации соответствующей оценки зависимости для аллель-взаимодействующих переменных. Каждая оценка зависимости сначала преобразуется при помощи функции ƒ(⋅) для генерации неявных вероятностей презентации на аллель  Вероятности на аллель
Вероятности на аллель 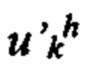 объединяются, и функция клиппирования может применяться к объединенным вероятностям для фиксации значений в диапазоне [0, 1], чтобы генерировать вероятность презентации того, что пептидная последовательность pk будет презентирована совокупностью ГКГС. Функция зависимости gh может быть в форме любой из функций зависимости gh, введенных выше в разделах VIII.B.1.
объединяются, и функция клиппирования может применяться к объединенным вероятностям для фиксации значений в диапазоне [0, 1], чтобы генерировать вероятность презентации того, что пептидная последовательность pk будет презентирована совокупностью ГКГС. Функция зависимости gh может быть в форме любой из функций зависимости gh, введенных выше в разделах VIII.B.1.
[00399] В качестве примера, вероятность того, что пептид pk будет презентирован аллелями ГКГС h=2, h=3„ среди m=4 различных идентифицированных аллелей ГКГС с использованием линейных функций преобразования gh(⋅), может быть получена путем:

где 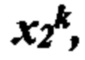
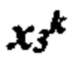 представляют собой идентифицированные аллель-взаимодействующие переменные для аллелей ГКГС h=2, h=3 и θ2, θ3 представляют собой совокупность параметров, определенных для аллелей ГКГС h=2, h=3.
представляют собой идентифицированные аллель-взаимодействующие переменные для аллелей ГКГС h=2, h=3 и θ2, θ3 представляют собой совокупность параметров, определенных для аллелей ГКГС h=2, h=3.
[00400] В качестве другого примера, вероятность того, что пептид pk будет презентирован аллелями ГКГС h=2, h=3, среди m=4 различных идентифицированных аллелей ГКГС с использованием сетевых функций преобразования gh(⋅), gw(⋅), может быть получена путем:
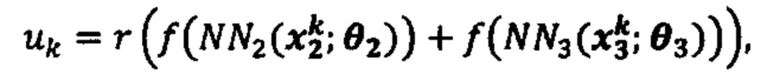
где NN2(⋅), представляют собой идентифицированные сетевые модели для аллелей ГКГС h=2, h=3 и θ2, θ3 представляют собой совокупность параметров, определяемых для аллелей ГКГС h=2, h=3.
[00401] На фиг. 11 изображено генерирование вероятности презентации для пептида pk в ассоциации с аллелями ГКГС h=2, h=3 с использованием иллюстративных сетевых моделей NN2(⋅) и NN3(⋅). Как изображено на фиг. 9, сетевая модель NN2(⋅) принимает аллель-взаимодействующие переменные 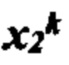 для аллеля ГКГС h=2 и генерирует выходные данные
для аллеля ГКГС h=2 и генерирует выходные данные 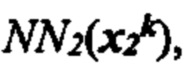 а сетевая модель NN3(⋅) принимает аллель-взаимодействующие переменные
а сетевая модель NN3(⋅) принимает аллель-взаимодействующие переменные 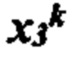 для аллеля ГКГС h=3 и генерирует выходные данные
для аллеля ГКГС h=3 и генерирует выходные данные 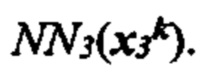 Каждый результат отображается функцией ƒ(⋅) и объединяется для генерации предполагаемой вероятности презентации uk.
Каждый результат отображается функцией ƒ(⋅) и объединяется для генерации предполагаемой вероятности презентации uk.
[00402] В другом варианте осуществления, если выполняются прогнозы для логарифма масс-спектрометрических ионных токов, r(⋅) представляет собой лог-функцию, а ƒ(⋅) представляет собой экспоненциальную функцию.
VIII.C.6. Пример 3.3: Модели суммы функций с аллель-невзаимодействующими переменными
[00403] В одном варианте осуществления неявная вероятность презентации на аллель для аллеля ГКГС h генерируется посредством:
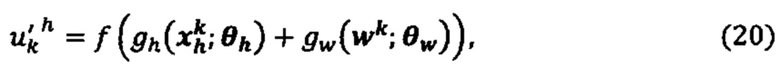
так что вероятность презентации получают путем:
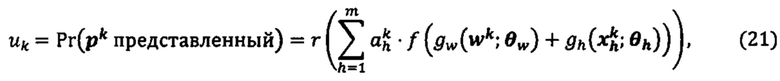
для включения влияния аллель-невзаимодействующих переменных на презентацию пептида.
[00404] Согласно уравнению (21) вероятность презентации того, что пептидная последовательность pk будет презентирована одним или более аллелями ГКГС H, может быть сгенерирована путем применения функции gh(⋅) к кодированной версии пептидной последовательности pk для каждого из аллелей ГКГС Н для генерации соответствующей оценки зависимости для аллель-взаимодействующих переменных для каждого аллеля ГКГС h. Функция gw(⋅) для аллель-невзаимодействующих переменных также применяется к кодированной версии аллель-невзаимодействующих переменных для генерации оценки зависимости для аллель-невзаимодействующих переменных. Оценка для аллель-невзаимодействующих переменных объединяется с каждым из оценок зависимости для аллель-взаимодействующих переменных. Каждый из комбинированных показателей преобразуется функцией _ƒ(⋅) для генерации неявных вероятностей презентации на аллель. Неявные вероятности объединяются, и функция клиппирования может применяться к объединенным выходным данным для фиксации значений в диапазоне [0,1], чтобы генерировать вероятность презентации того, что пептидная последовательность pk будет презентирована аллелями ГКГС Н. Функция зависимости gw может быть в формме любой из функций зависимости gw, введенных выше в разделах VIII.B.3.
[00405] В качестве примера, вероятность того, что пептид pk будет презентирован аллелями ГКГС h=2, h=3, среди m=4 различных идентифицированных аллелей ГКГС с использованием линейных функций преобразования gh(⋅), gw(⋅), может быть получена путем:
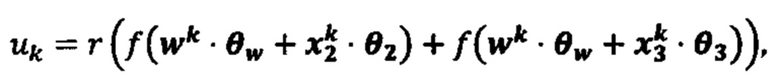
где wk представляют собой идентифицированные аллель-невзаимодействующие переменные для пептида pk, и θw представляют собой совокупность параметров, определенных для аллель-невзаимодействующих переменных.
[00406] В качестве другого примера, вероятность того, что пептид pk будет презентирован аллелями ГКГС h=2, h=3, среди m=4 различных идентифицированных аллелей ГКГС с использованием сетевых функций преобразования gh(⋅), gw(⋅), может быть получена путем:
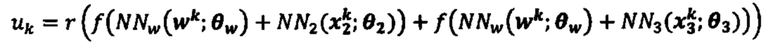
где wk представляют собой идентифицированные аллель-невзаимодействующие переменные для пептида pk, и θw представляют собой совокупность параметров, определенных для аллель-невзаимодействующих переменных.
[00407] На фиг. 12 изображено генерирование вероятности презентации для пептида pk в ассоциации с аллелями ГКГС h=2, h=3 с использованием иллюстративных сетевых моделей NN2(⋅), NN3(⋅), и NNw(⋅). Как изображено на фиг. 12, сетевая модель NN2(⋅) принимает аллель-взаимодействующие переменные 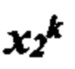 для аллеля ГКГС h=2 и генерирует выходные данные
для аллеля ГКГС h=2 и генерирует выходные данные 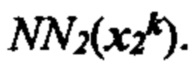 Сетевая модель NNw(⋅) принимает аллель-невзаимодействующие переменные wk для пептида pk и генерирует выходные данные NNw(wk). Результаты комбинируются и отображаются при помощи функции ƒ(⋅). Сетевая модель NN3(⋅) принимает аллель-взаимодействующие переменные
Сетевая модель NNw(⋅) принимает аллель-невзаимодействующие переменные wk для пептида pk и генерирует выходные данные NNw(wk). Результаты комбинируются и отображаются при помощи функции ƒ(⋅). Сетевая модель NN3(⋅) принимает аллель-взаимодействующие переменные 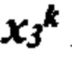 для аллеля ГКГС h=3 и генерирует выходные данные
для аллеля ГКГС h=3 и генерирует выходные данные 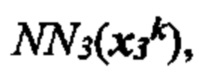 которые снова объединяют с выходными данными NNw(wk) той же сетевой модели NNw(⋅) и отображаемые функцией ƒ(⋅). Оба результата объединяют для генерации предполагаемой вероятности презентации uk.
которые снова объединяют с выходными данными NNw(wk) той же сетевой модели NNw(⋅) и отображаемые функцией ƒ(⋅). Оба результата объединяют для генерации предполагаемой вероятности презентации uk.
[00408] В другом варианте осуществления неявная вероятность презентации на аллель для аллеля ГКГС h генерируется посредством:
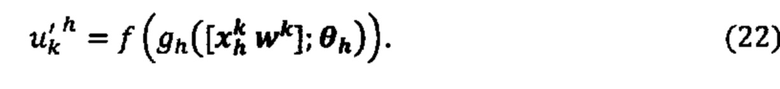
так что вероятность презентации получают путем:
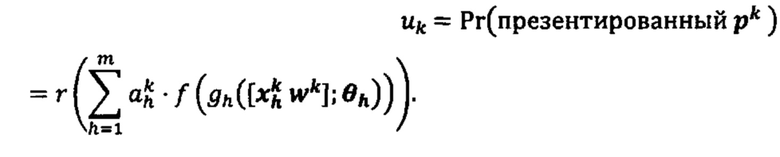
VIII.C.7. Пример 4: Модели второго порядка
[00409] В одном варианте осуществления s(⋅) является функцией второго порядка, а предполагаемая вероятность презентации uk для пептида pk определяется следующим образом:
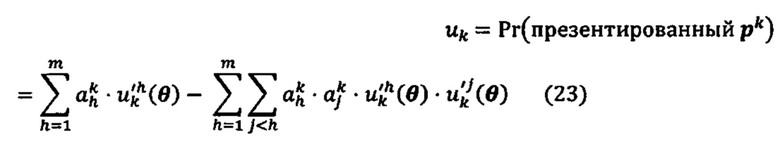
где элементы 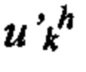 представляют собой неявную вероятность презентации на аллель для аллеля ГКГС h. Значения для совокупности параметров θ для неявных вероятностей на аллель могут быть определены путем минимизации функции потерь по θ, где i представляет собой каждый объект в подсовокупности S данных для обучения 170, генерируемых из клеток, экспрессирующих одиночные аллели ГКГС, и/или клеток, экспрессирующих множественные аллели ГКГС. Неявные вероятности презентации для каждого аллеля могут быть в любой форме, показанной в уравнениях (18), (20) и (22), описанных выше.
представляют собой неявную вероятность презентации на аллель для аллеля ГКГС h. Значения для совокупности параметров θ для неявных вероятностей на аллель могут быть определены путем минимизации функции потерь по θ, где i представляет собой каждый объект в подсовокупности S данных для обучения 170, генерируемых из клеток, экспрессирующих одиночные аллели ГКГС, и/или клеток, экспрессирующих множественные аллели ГКГС. Неявные вероятности презентации для каждого аллеля могут быть в любой форме, показанной в уравнениях (18), (20) и (22), описанных выше.
[00410] В одном аспекте модель уравнения (23) может означать, что существует вероятность того, что пептид pk будет презентирован двумя аллелями ГКГС одновременно, в которой презентация двумя аллелями HLA является статистически независимым.
[00411] Согласно уравнению (23) вероятность презентации того, что пептидная последовательность pk будет презентирована одним или более аллелями ГКСГ Н, может быть сгенерирована путем комбинирования неявных вероятностей презентации для каждого аллеля и вычитания вероятности того, что каждая пара аллелей ГКГС будет одновременно презентировать пептид pk из суммы для генерации вероятности презентации того, что пептидная последовательность pk будет презентирована аллелями ГКГС Н.
[00412] В качестве примера, вероятность того, что пептид pk будет презентирован аллелями HLA h=2, h=3, среди m=4 различных идентифицированных аллелей HLA с использованием линейных функций преобразования gh(⋅), может быть получена путем:
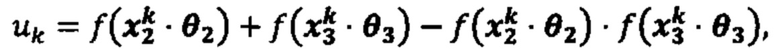
где  представляют собой идентифицированные аллель-взаимодействующие переменные для аллелей HLA h=2, h=3 и θ2, θ3 представляет собой совокупность параметров, определенных для аллелей HLA h=2, h=3.
представляют собой идентифицированные аллель-взаимодействующие переменные для аллелей HLA h=2, h=3 и θ2, θ3 представляет собой совокупность параметров, определенных для аллелей HLA h=2, h=3.
[00413] В качестве другого примера, вероятность того, что пептид рk будет презентирован аллелями HLA h=2, h=3, среди m=4 различных идентифицированных аллелей HLA с использованием сетевых функций преобразования gh(⋅), gw(⋅), может быть получена путем:
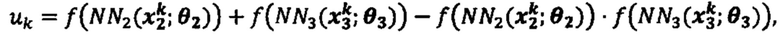
где NN2(⋅), NN3(⋅) представляют собой идентифицированные сетевые модели для аллелей HLA h=2, h=3 и θ2, θ3 представляют собой совокупность параметров, определяемых для аллелей HLA А=2, h=3.
IX. Пример 5: Модуль прогнозирования
[00414] Модуль прогнозирования 320 принимает данные последовательности и выбирает кандидатные неоантигены в данных последовательности с использованием моделей презентации. В частности, данные последовательности могут быть последовательностями ДНК, последовательностями РНК и/или белковыми последовательностями, выделенными из клеток опухолевых тканей пациентов. Модуль прогнозирования 320 обрабатывает данные последовательности во множество пептидных последовательностей pk, имеющих 8-15 аминокислот. Например, модуль прогнозирования 320 может обрабатывать данную последовательность «IEFROEIFJEF» в три пептидные последовательности, содержащие 9 аминокислот "IEFROEIFJ," "EFROEIFJE," и "FROEIFJEF." В одном варианте осуществления модуль прогнозирования 320 может идентифицировать кандидатные неоантигены, которые являются мутантными пептидными последовательностями, путем сравнения данных последовательности, извлеченных из нормальных тканевых клеток пациента, с данными последовательности, извлеченными из клеток опухолевой ткани пациента, для идентификации участков, содержащих одну или более мутаций.
[00415] Модуль презентации 320 применяет одну или более моделей презентации к обрабатываемым пептидным последовательностям для оценки вероятности презентации пептидных последовательностей. В частности, модуль прогнозирования 320 может выбирать одну или более пептидных последовательностей кандидатных неоантигенов, которые имеют вероятность быть презентированными на опухолевых молекулах HLA, путем применения модели презентации к кандидатным неоантигенам. В одном варианте осуществления модуль презентации 320 выбирает последовательности кандидатных неоантигенов, которые имеют предполагаемые вероятности презентации выше заданного порогового значения. В другом варианте осущестления модель презентации выбирает N последовательностей кандидатных неоантигенов, которые имеют наивысшие предполагаемые вероятности презентации (где N, как правило, представляет собой максимальное количество эпитопов, которые могут быть доставлены в вакцину). Вакцина, содержащая выбранные кандидатные неоантигены для данного пациента, может быть введена пациенту для индуцирования иммунных реакций.
X. Пример 6: Результаты эксперимента, демонстрирующие эффективность иллюстративной модели презентации
[00416] Пригодность различных моделей презентации, описанных выше, была проверена на тестовых данных Т, которые представляли собой подмножествами данных для обучения 170, которые не использовались для обучения моделей презентации или отдельной совокупности данных из данных для обучения 170, которые имеют аналогичные переменные и структуры данных в виде данных для обучения 170.
[00417] Соответствующим показателем эффективности моделей презентации является: Прогностическая ценность положительного результата 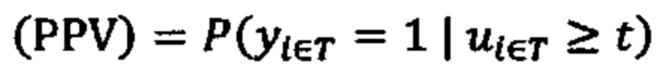
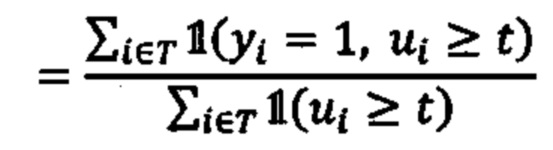
что указывает на отношение числа объектов пептида, презентированных на HLA-ассоциированных аллелях, которые были правильно спрогнозированы к числу объектов пептида, которые, как прогнозировалось, были презентированы на аллелях HLA. В одном варианте осуществления было спрогнозировано, что пептид pi в тестовых данных Т презентирован на одном или более HLA-ассоциированных аллелях, если соответствующая оценка вероятности ui больше или равна заданному пороговому значению t. Соответствующим показателем эффективности моделей презентации является:
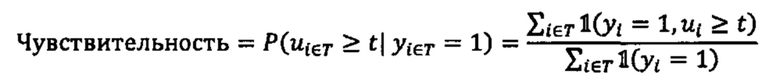
что указывает на отношение числа объектов пептида, которые были правильно спрогнозированы, чтобы быть презентированными на HLA-ассоциированных аллелях к числу объектов пептида, которые, как известно, были презентированы на аллелях HLA. Соответствующим показателем эффективности моделей презентации является площадь под кривой (AUC) зависимости доли истинно положительных от доли ложно положительных результатов (ROC). График ROC отображает чувствительность против доли ложно положительных результатов (FPR), которая определяется:

Х.А. Сравнение эффективности модели презентации по данным масс-спектрометрии против современной модели
[00418] На фиг. 13А сравниваются результаты тестирования эффективности иллюстративной модели презентации, представленной в данном документе, и современных моделей для прогнозирования презентации пептида по данным масс-спектрометрии множества аллелей. Результаты продемонстрировали, что иллюстративная модель презентации значительно улучшилась в прогнозировании презентации пептидов по сравнению с современными моделями, основанными на прогнозировании аффинности и стабильности.
[00419] В частности, иллюстративная модель презентации, изображенная на фиг. 13А как «MS», представляла собой максимум модели презентации для каждого аллеля, показанной в уравнении (12) с использованием линейной функции зависимости gh(⋅) и логистической функции _ƒ(⋅). Иллюстративная модель презентации была обучена на основе подсовокупности данных масс-спектрометрии одного аллеля HLA-A*02:01 из совокупности данных IEDB (совокупность данных «D1») (данные можно найти по адресу http://www.iedb.org/doc/mhc_ligand_full.zip) и подсовокупности данных масс-спектрометрии одного аллеля HLA-B *07:02 из совокупности данных IEDB (совокупность данных «D2») (данные можно найти по адресу http://www.iedb.org/doc/mhc_ligand_full.zip). Все пептиды из исходного белка, которые содержат презентированные пептиды в тестовой совокупности, были исключены из данных для обучения, так что иллюстративная модель презентации не могла просто запомнить последовательности презентированных антигенов.
[00420] Модель изображена на фиг. 13А как «аффинность» представляла собой модель, подобную современной модели, которая прогнозирует презентацию пептида на основе прогнозирования аффинности NETMHCpan. Практическое применение NETMHCpan подробно описано на http://www.cbs.dtu.dk/services/NetMHCpan/. Модель, изображенная на фиг. 13А, как «стабильность», представляла собой модель, подобную современной модели, которая прогнозирует презентацию пептида на основе прогнозирования устойчивости NETMHCstab. Практическое применение NETMHCstab подробно описано на http://www.cbs.dtu.dk/services/NetMHCstab-l.0/. Тестовые данные, которые являются подсовокупностью данных масс-спектрометрии многоаллельной JY-клеточной линии HLA-A*02:01 и HLA-B*07:02 из совокупности данных Бассани-Штернберг (совокупность данных «D3») (данные можно найти по адресу: www.ebi.ac.uk/pride/archive/projects/PXD000394). Планки погрешностей (как показано сплошными линиями) демонстрируют 95% доверительные интервалы.
[00421] Как показано на фиг. 13А иллюстративная модель презентации, прошедшая обучение по данным масс-спектрометрии, имела значительно более высокое значение PPV с 10% чувствительностью по сравнению с современными моделями, которые прогнозируют презентацию пептида на основе прогнозов аффинности связывания ГКГС или прогнозов стабильности связывания ГКГС. В частности, иллюстративная модель презентации имела приблизительно на 14% выше PPV, чем модель, основанная на спрогнозированных аффинности, и имела приблизительно на 12% выше PPV, чем модель, основанная на прогнозах стабильности.
[00422] Эти результаты демонстрируют, что иллюстративная модель презентации имела значительно лучшую эффективность, чем современные модели, которые прогнозируют презентацию пептида на основе аффинности связывания ГКГС или стабильности связывания ГКГС, хотя иллюстративная модель презентации не была обучена на основе белковых последовательностей, которые содержали презентированные пептиды.
Х.В. Сравнение эффективности модели презентации по данным эпитопов Т-клеток против современных моделей
[00423] На фиг. 13В сравниваются результаты тестирования эффективности иллюстративной модели презентации, представленной в данном документе, и современных моделей для прогнозирования презентации пептида по данным эпитопов Т-клеток. Данные эпитопов Т-клеток содержат пептидные последовательности, которые были презентированы аллелями ГКГС на поверхности клеток и распознаются Т-клетками. Результаты показали, что, хотя иллюстративная модель презентации обучена на основе данных масс-спектрометрии, иллюстративная модель презентации значительно улучшилась при прогнозировании Т-клеточных эпитопов по сравнению с современными моделями, основанные на прогнозах аффинности и стабильности. Другими словами, результатами на фиг. 13В показано, что иллюстративная модель презентации не только лучше, чем современные модели при прогнозировании презентации пептида по тестовым данным масс-спектрометрии, но иллюстративная модель презентации также выполнялась значительно лучше, чем современные модели при прогнозировании эпитопов, которые были фактически распознаны Т-клетками. Это указывает на то, что разнообразие моделей презентации, представленных в данном документе, может обеспечить улучшенную идентификацию антигенов, которые могут индуцировать иммуногенные реакции в иммунной системе.
[00424] В частности, иллюстративная модель презентации, изображенная на фиг. 13В как «MS», представляла собой модель презентации для каждого аллеля, показанную в уравнении (2), с использованием линейной функции преобразования gh(⋅) и логистической функции ƒ(⋅), которая была обучена на основе подсовокупности из совокупности данных D1. Все пептиды из исходного белка, которые содержат презентированные пептиды в тестовой совокупности, были исключены из данных для обучения, так что модель презентации не могла просто запомнить последовательности презентированных антигенов.
[00425] Каждая из моделей была применена к тестовым данным, которые являются подсовокупностью данных масс-спектрометрии по данным эпитопов Т-клеток HLA-А*02:01 (совокупность данных «D4») (данные можно найти на сайте www.iedb.org/doc/tcell full v3.zip). Модель, изображенная на фиг. 13В как «аффинность» представляла собой модель, подобную современной модели, которая прогнозирует презентацию пептида на основе прогнозов аффинности NETMHCpan, и модель, изображенная на фиг. 13В, как «стабильность», представляла собой модель, подобную современной модели, которая прогнозирует презентацию пептида на основе прогнозов устойчивости NETMHCstab. Планки погрешностей (как показано сплошными линиями) демонстрируют 95% доверительные интервалы.
[00426] Как показано на фиг. 13А, модель презентации каждого аллеля, подготовленная по данным масс-спектрометрии, имела значительно более высокое значение PPV с 10% чувствительностью, чем современные модели, которые прогнозируют презентацию пептидов на основе аффинности связывания ГКГС или стабильности связывания ГКГС, даже если модель презентации не была обучена на основе белковых последовательностей, которые содержали презентированные пептиды. В частности, модель презентации для каждого аллеля имела приблизительно на 9% выше PPV, чем модель, основанная на прогнозах аффинности, и имела приблизительно на 8% выше PPV, чем модель, основанная на прогнозах стабильности.
[00427] Эти результаты показали, что иллюстративная модель презентации, обученная на данных масс-спектрометрии, была значительно лучше, чем современные модели прогнозирования эпитопов, которые были распознаны Т-клетками.
Х.С. Сравнение эффективности различных моделей по данным масс-спектрометрии
[00428] На фиг. 13С сравниваются результаты тестирования эффективности для иллюстративной модели функции сумм (уравнение (13)), иллюстративной модели суммы функций (уравнение (19)) и иллюстративной модели второго порядка (уравнение (23)) для прогнозирования презентации пептида по данным масс-спектрометрии множества аллелей. Результаты показали, что модель суммы функций и модель второго порядка лучше, чем модель функции сумм. Это связано с тем, что модель функции сумм подразумевает, что аллели при многоаллельных параметрах могут мешать друг другу для презентации пептидов, когда на самом деле презентация пептидов эффективна независимо.
[00429] В частности, иллюстративная модель презентации, обозначенная как «сигмоид сумм» на фиг. 13С, представляла собой модель функции сумм с использованием сетевой функции зависимости gh(⋅), функции тождественности ƒ(⋅), и логистической функции r(⋅). Иллюстративная модель, обозначенная как «сумма сигмоидов», представляла собой модель суммы функций в уравнении (19) с сетевой функцией зависимости gh(⋅), логистической функцией ƒ(⋅), и функцией тождественности r(⋅). Иллюстративная модель, обозначенная как «гиперболический тангенс», представляла собой модель суммы функций в уравнении (19) с сетевой функцией зависимости gh(⋅), логистической функцией ƒ(⋅), и функцией гиперболического тангенса r(⋅). Иллюстративная модель, обозначенная как «второй порядок», была моделью второго порядка в уравнении (23) с использованием формы неявных вероятностей презентации для каждого аллеля, определенной в уравнении (18), с сетевой функцией зависимости gh(⋅) и логистической функцией ƒ(⋅). Каждая модель была обучена на основе подсовокупности из совокупности данных D1, D2 и D3. Примеры моделей презентации были применены к тестовым данным, которые являются случайной подсовокупностью из совокупности данных D3, которая не совпадает с данными для обучения.
[00430] Как изображено на фиг. 13С, первый столбец относится к AUC ROC, когда каждая модель презентации была применена к тестовой совокупности, второй столбец относится к значению потери отрицательной логарифмической функции правдоподобия, а третья колонка относится к PPV с 10% чувствительностью. Как изображено на фиг. 13С, эффективность моделей презентации «сумма сигмоидов», «гиперболический тангенс» и «второй порядок» были приблизительно привязаны к приблизительно 15-16% PPV при 10%-ной чувствительности, тогда как эффективность модели «сигмоид сумм» была немного ниже приблизительно 11%.
[00431] Как обсуждалось ранее в разделе VIII.C.4, результаты показали, что модели презентации «сумма сигмоидов», «гиперболический тангенс» и «второй порядок» имеют высокие значения PPV по сравнению с «сигмоидом суммы", потому что модели правильно учитывают, что пептиды презентированы независимо каждым аллелем ГКГС в условиях множества аллелей.
X.D. Сравнение эффективности модели презентации с обучением и без обучения по данным масс-спектрометрии одного аллеля
[00432] На фиг. 13D сравниваются результаты тестирования эффективности для двух иллюстративных моделей презентации, которые обучаются с и без данных масс-спектрометрии одного аллеля для прогнозирования презентации пептида для данных масс-спектрометрии множества аллелей. Результаты показали, что иллюстративные модели презентации, которые прошли обучение без данных одного аллеля, достигают сопоставимых результатов с примерами моделей презентации, обученных с использованием данных одного аллеля.
[00433] Иллюстративная модель «с данными одного аллеля А2/В7» была моделью презентации «сумм сигмоидов» в уравнении (19) с сетевой функцией зависимости gh(⋅), логистической функцией ƒ(⋅), и функцией тождественности r(⋅). Модель была обучена на основе подсовокупности из совокупности данных D3 и данных масс-спектрометрии одного аллеля для множества аллелей ГКГС из базы данных IEDB (данные можно найти по адресу: http://www.iedb.org/doc/mhc_ligand_full.zip). Иллюстративная модель «без данных одного аллеля А2/В7» была одной и той же моделью, но была обучена на основе подсовокупности из совокупности данных множества аллелей D3 без данных масс-спектрометрии одного аллеля для аллелей HLA-A *02:01 и HLA -В*07:02, но с данными масс-спектрометрии одного аллелля для других аллелей. В многоаллельных данных для обучения, клеточная линия НСС1937 экспрессировала HLA-B*07:02, но не HLA-A*02:01, а клеточная линия НСТ116 экспрессировала HLA-A*02: 01, но не HLA-B*07:02. Примеры моделей презентации были применены к тестовым данным, которые были случайной подсовокупностью из совокупности данных D3 и не совпадали с данными для обучения.
[00434] Столбец «Корреляция» относится к корреляции между фактическими метками, которые указывают, был ли пептид презентирован на соответствующем аллеле в тестовых данных, и меткой для прогнозирования. Как изображено на фиг. 13D, прогнозы, основанные на неявной вероятности презентации для каждого аллеля для аллеля ГКГС HLA-A* 02:01, выполнялись значительно лучше на тестовых данных одного аллеля для аллеля ГКГС HLA-А*02: 01, чем для аллеля ГКГС HLA-B*07:02. Аналогичные результаты показаны для аллеля ГКГС HLA-B*07:02.
[00435] Эти результаты указывают на то, что неявные вероятности презентации для каждого аллеля модели презентации могут правильно прогнозировать и различать связывающие мотивы к отдельным аллелям ГКГС, даже если прямая связь между пептидами и каждым отдельным аллелем ГКГС не была известна в данных для обучения.
Х.Е. Сравнение эффективности прогнозирования для каждого аллеля без обучения по данным масс-спектрометрии одного аллеля
[00436] На фиг. 13Е изображена эффективность для моделей «без данных одного аллеля А2/В7» и «с использованием данных одного аллеля А2/В7», изображенных на фиг. 13D, по данным масс-спектрометрии одного аллеля для аллелей HLA-A*02:01 и HLA-B*07:02, которые были проведены в анализе, изображенном на фиг. 13D. Результаты демонстрируют, что даже через иллюстративную модель презентации обучают без данных масс-спектрометрии одного аллеля для этих двух аллелей, модель способна изучать связывающие мотивы для каждого аллеля ГКГС.
[00437] Как изображено на фиг. 13Е, «модель А2, прогнозирующая В7», указывает на эффективность модели, когда презентация пептида прогнозируется для данных одного аллеля HLA-B*07: 02 на основе неявной вероятности презентации для каждого аллеля, оценивающей аллель ГКГС HLA-A*02:01. Аналогично, «модель А2, прогнозирующая А2», указывает на эффективность модели, когда презентация пептида прогнозируется для одного аллеля HLA-A*02:01 на основе неявной оценки вероятности презентации для каждого аллеля для аллелей ГКГС HLA-A*02:01. «Модель В7, прогнозирующая В7», указывает на эффективность модели, когда презентация пептида прогнозируется для данных одного аллеля HLA-B*07:02 на основе неявной вероятности презентации для каждого аллеля, оценивающей аллель ГКГС HLA-B*07:02. «Модель В7, прогнозирующая А2», указывает на эффективность модели, когда презентация пептида прогнозируется для одного аллеля HLA-А*02:01 на основе неявной оценки вероятности презентации для каждого аллеля для аллелей ГКГС HLA-B*07:02.
[00438] Как изображено на фиг. 13Е, прогностическая способность неявных вероятностей для каждого аллеля для аллеля HLA значительно выше для предполагаемого аллеля и значительно ниже для другого аллеля HLA. Аналогично результатам, изображенным на фиг. 13D, иллюстративные модели презентации правильно обучили для различения презентации пептидов отдельных аллелей HLA-A*02:01 и HLA-B*07: 02, хотя прямая связь между презентацией пептидов и этими аллелями отсутствовала в многоаллельных данных для обучения.
X.F. Часто повторяющиеся якорные остатки в прогнозах на аллель соответствуют известным каноническим якорным мотивам
[00439] На фиг. 13F показаны общие якорные остатки в положениях 2 и 9 среди полимеров из девяти мономерных звеньев, спрогнозированным при помощи модели «без данных одного аллеля А2/В7», показанной на фиг. 13D. Предполагалось, что пептиды будут презентированы, если предполагаемая вероятность будет выше 5%. Результаты демонстрируют, что наиболее распространенные якорные остатки в пептидах, идентифицированных для презентации на аллелях ГКГС HLA-A*02:01 и HLA-B*07:02, соответствуют ранее известным якорным мотивам для этих аллелей ГКГС. Это указывает на то, что иллюстративные модели презентации правильно изучили пептидное связывание, основанное на определенных положениях аминокислот пептидных последовательностей, как и ожидалось.
[00440] Как изображено на фиг. 13F, аминокислоты L/M в положении 2 и аминокислоты V/L в положении 9 были известны как мотивы канонических якорных остатков (как показано в таблице 4 из https://link.springer.com/article/10.1186/1745-7580-4-2) для HLA-А*02:01 и аминокислоты Р в положении 2 и аминокислоты L/V в положении 9, как известно, являются мотивом канонических якорных остатков для HLA-B*07:02. Наиболее распространенные мотивы якорных остатков в положениях 2 и 9 для пептидов идентифицировали модель, соответствующую моделям известных мотивов канонических якорных остатков для обоих аллелей HLA.
X.G. Сравнение эффективности модели презентации с и без аллель-невзаимодействующих переменных
[00441] На фиг. 13G сравниваются результаты тестирования эффективности между иллюстративной моделью презентации, которая включала С- и N-концевые фланкирующие последовательности в качестве аллель-взаимодействующих переменных, и иллюстративной моделью презентации, которая включала С- и N-концевые фланкирующие последовательности как аллель-невзаимодействующие переменные. Результаты показали, что включение С- и N-концевых фланкирующих последовательностей в качестве аллель-невзаимодействующих переменных значительно улучшает эффективность модели. Более конкретно, полезно идентифицировать соответствующие признаки для презентации пептидов, которые являются общими для разных аллелей ГКГС, и моделировать их таким образом, чтобы статистическая сила для этих аллель-невзаимодействующих переменных была распределена по аллелям ГКГС для улучшения эффективности модели презентации.
[00442] Иллюстративная «аллель-взаимодействующая» модель была моделью суммы функций, использующая форму неявных вероятностей презентации для каждого аллеля в уравнении (22), которые включали С- и N-концевые фланкирующие последовательности в качестве аллель-взаимодействующих переменных, с сетевой функцией зависимости gh(⋅) и логистической функцией ƒ(⋅). Иллюстративная «аллель-взаимодействующая» модель была моделью суммы функций, показанной в уравнении (21), которая включала С- и N-концевые фланкирующие последовательности в качестве аллель-невзаимодействующих переменных, с сетевой функцией зависимости gh(⋅) и логистической функцией ƒ(⋅). Аллель-невзаимодействующие переменные были смоделированы с помощью отдельной сетевой функции зависимости gw(⋅). Обе модели были обучены на подсовокупности из совокупности данных D3 и данных масс-спектрометрии одного аллеля для множества аллелей ГКГС из базы данных IEDB (данные можно найти по адресу: http://www.iedb.org/doc/mhc_ligand_full.zip). Каждую из моделей презентации применяли к совокупности тестовых данных, которая является случайной подсовокупностью из совокупности данных D3, которая не совпадает с данными для обучения.
[00443] Как изображено на фиг. 13G, включение С- и N-концевые фланкирующих последовательностей в иллюстративную модель презентации как аллель-невзаимодействующих переменных обеспечивало приблизительно 3%-ное улучшение значения PPV относительно моделирования их как аллель-взаимодействующих переменных. Это связано с тем, что в общем случае «аллель-невзаимодействующая» иллюстративная модель презентации способна распределить статистическую мощность аллель-невзаимодействующих переменных в аллелях ГКГС путем моделирования эффекта с помощью отдельной сетевой функции зависимости с очень небольшим добавлением вычислительной мощности.
Х.Н. Зависимость между презентированными пептидами и количественной оценкой мРНК
[00444] На фиг. 13Н изображена зависимость между фракцией презентированных пептидов для генов на основе количественной оценки мРНК для масс-спектрометрических данных опухолевых клеток. Результаты демонстрируют, что существует сильная зависимость между экспрессией мРНК и презентацией пептида.
[00445] В частности, горизонтальная ось на фиг. 13G указывает на экспрессию мРНК в пересчете на квартили транскриптов на миллион (ТРМ). Вертикальная ось на фиг. 13G обозначает фракцию презентированных эпитопов из генов в соответствующих квартилях экспрессии мРНК. Каждая сплошная линия представляет собой график зависимости двух измерений от образца опухоли, который связан с соответствующими данными масс-спектрометрии и измерениями экспрессии мРНК. Как изображено на фиг. 13G, наблюдается сильная положительная корреляция между экспрессией мРНК и фракцией пептидов в соответствующем гене. В частности, пептиды из генов в верхнем квартиле экспрессии РНК более чем в 20 раз, вероятно, будут презентированы, чем в нижнем квартиле. Кроме того, по существу 0 пептидов презентированы из генов, которые не обнаруживаются посредством РНК.
[00446] Результаты демонстрируют, что эффективность модели презентации может быть значительно улучшена за счет включения количественных измерений мРНК, поскольку эти измерения являются сильными прогностическим фактором презентации пептида.
X.I. Сравнение эффективности модели презентации с включением данных количественной оценки РНК
[00447] На фиг. 13I изображена эффективность двух иллюстративных моделей презентации, одна из которых обучается на основе данных масс-спектрометрии опухолевых клеток, другая из которых содержит данные количественной оценки мРНК и данные масс-спектрометрии опухолевых клеток. Как и ожидалось на фиг. 13Н, результаты показали, что есть существенное улучшение эффективности путем включения количественных измерений мРНК в иллюстративную модель презентации, поскольку экспрессия мРНК является сильным индикатором презентации пептида.
[00448] «MHCflurry + РНК-фильтр» представляла собой модель, подобную современной модели, которая прогнозирует презентацию пептида на основе прогнозов аффинности. Она был реализована с использованием NHCflurry вместе со стандартным фильтром экспрессии гена(ов), который удалял все пептиды из белков с помощью количественных измерений мРНК, которые составляли менее 3,2 FPKM. Реализация MHCflyrry подробно описана на сайте https://github.com/hammerlab/mhcflurry/. и на сайте http://biorxiv.org/content/early/2016/05/22/054775. Модель «иллюстративная модель, без РНК» представляла собой иллюстративную модель презентации «сумма сигмоидов», представленную в уравнении (21), с сетевой функцией зависимости gh(⋅), сетевой функцией зависимости gw(⋅), и логистической функцией ƒ(⋅). Модель «иллюстративная модель, без РНК» включала С-концевые фланкирующие последовательности как аллель-невзаимодействующие переменные через сетевую функцию зависимости gw(⋅).
[00449] Модель «иллюстративная модель с РНК» представляла собой модель презентации «сумма сигмоидов», представленную в уравнении (19) с сетевой функцией зависимости gh(⋅), сетевой функцией зависимости gw(⋅) в уравнении (10), включающую данные количественной оценки мРНК через лог-функцию и логистическую функцию ƒ(⋅). Модель «иллюстративная модель с РНК» включала С-концевые фланкирующие последовательности в качестве аллель-невзаимодействующих переменных через сетевую функцию зависимости gw(⋅) и включала количественные измерения мРНК через лог-функцию.
[00450] Каждая модель была обучена на комбинации данных масс-спектрометрии одного аллеля из совокупности данных IEDB, 7 клеточных линий из данных масс-спектрометрии множества аллелей из совокупности данных Бассани-Штернберга и масс-спектрометрии 20 образцов опухоли. Каждая модель была применена к тестовой совокупности, включающей 5000 удерживаемых белков из 7 образцов опухолей, которые составляли 9830 презентированных пептидов из общего числа 52156840 пептидов.
[00451] Как изображено на первых двух гистограммах фиг. 13I, модель «Иллюстративная модель без РНК» имеет значение PPV 21% при 20% чувствительности, а у современной модели - около 3%. Это указывает на начальное улучшение эффективности на 18% в значении PPV, даже без учета количественных измерений мРНК. Как изображено на третьей диаграмме фиг. 13I, модель «иллюстративная модель с РНК», которая включает данные количественной оценки мРНК в модели презентации, демонстрирует значение PPV около 30%, что почти на 10% выше по сравнению с иллюстративной моделью презентации без количественных измерений мРНК.
[00452] Таким образом, результаты демонстрируют, что, как и ожидалось из результатов на фиг. 13Н, экспрессия мРНК действительно является сильным прогностическим фактором для прогнозирования пептидов, что позволяет значительно улучшить эффективность модели презентации с очень небольшим добавлением вычислительной сложности.
X.J. Пример параметров, определенных для аллеля ГКГС HLA-C*16:04
[00453] На фиг. 13J сравнивается вероятность презентации пептида для разных длин пептидов между результатами, полученными при помощи модели презентации «иллюстративная модель с РНК», описанной со ссылкой на фиг. 13I, и спрогнозированными результатами при помощи современных моделей, которые не учитывают длину пептида при прогнозировании презентации пептида. Результаты показали, что иллюстративная модель презентации «иллюстративная модель с РНК» из фиг. 13I выявила вариации в вероятностях по всем пептидам различной длины.
[00454] Горизонтальная ось обозначала образцы пептидов длиной 8, 9, 10 и 11. Вертикальная ось обозначала вероятность презентации пептида, обусловленного длинами пептида. На графике «Фактическая вероятность, основанная на тестовых данных» была показана доля презентированных пептидов в зависимости от длины пептида в совокупности тестовых данных. Вероятность презентации варьировала в зависимости от длины пептида. Например, как изображено на фиг. 13J, 10-мерный пептид с каноническими HLA-A2 L/V якорными мотивами был примерно в 3 раза менее вероятным, чем 9-мерный с теми же якорными остатками. График «Модели, игнорирующие длину», указывал на спрогнозированные измерения, если современные модели, которые игнорируют длину пептида, должны были применяться к той же совокупности тестовых данных для прогнозирования презентации. Эти модели могут быть версиями NetMHC до версии 4.0, NetMHCpan до версии 3.0 и MHCflurry, которые не учитывают изменения в презентации пептида в соответствии с длиной пептида. Как изображено на фиг. 13J, доля презентированных пептидов была бы постоянной при разных значениях длины пептида, что указывает на то, что эти модели не могут выявить вариации в презентации пептида в соответствии с длиной. График «Gritston с РНК» показал измерения, полученные из модели презентации «Gritstone с РНК». Как изображено на фиг. 13J, измерения, сгенерированные при помощи модели «Gritstone с РНК», внимательно следили за показателями, показанными в «Фактической вероятности тестовых данных», и правильно учитывали разную степень презентации пептидов для длин 8, 9, 10 и 11.
[00455] Таким образом, результаты показали, что представленные в данном документе иллюстративные модели презентации приводили к улучшению прогнозов не только для 9-мерных пептидов, но и для пептидов другой длины от 8 до 15, на которые приходится до 40% презентированных пептидов в аллелях класса IHLA.
Х.K. Пример параметров, определенных для аллеля ГКГС HLA-C* 16:04
[00456] Ниже показан совокупность параметров, определенных для вариации модели презентации для каждого аллеля (уравнение (2)) для аллеля ГКСГК HLA-C*16: 04, обозначаемого при помощи h:
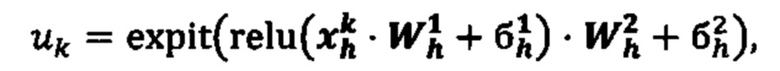
где relu (⋅) представляет собой функцию блока линейной ректификации (RELU), а 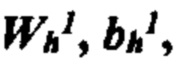
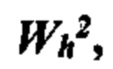 и
и 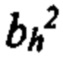 представляют собой совокупность параметров θ, определенных для модели. Аллель-взаимодействующие переменные
представляют собой совокупность параметров θ, определенных для модели. Аллель-взаимодействующие переменные 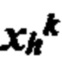 состоят из пептидных последовательностей. Размеры I
состоят из пептидных последовательностей. Размеры I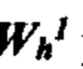 равны (231×256), размеры
равны (231×256), размеры 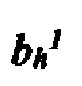 равны (1×256), размеры
равны (1×256), размеры 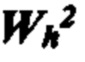 равны (256×1), а
равны (256×1), а 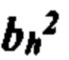 - скаляр. Для демонстрационных целей значения для
- скаляр. Для демонстрационных целей значения для 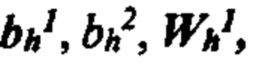 и
и 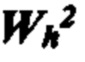 приведены ниже.
приведены ниже. 


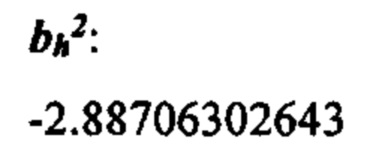


XI. Пример компьютера
[00457] На фиг. 14 изображен пример компьютера 1400 для имплементации объектов, изображенных на фиг. 1 и 3. Компьютер 1400 содержит по меньшей мере один процессор 1402, связанный с чипсетом 1404. Чипсет 1404 содержит контроллер-концентратор памяти 1420 и контроллер-концентратор ввода/вывода (I/O) 1422. Память 1406 и графический адаптер 1412 соединены с контроллером-концентратором памяти 1420, и дисплей 1418 соединен с графическим адаптером 1412. Устройство хранения данных 1408, устройство ввода 1414 и сетевой адаптер 1416 соединены с контроллером-концентратором ввода/вывода 1422. Другие варианты осуществления компьютера 1400 имеют разные архитектуры.
[00458] Устройство хранения данных 1408 является энергонезависимым машиночитаемым носителем данных, таким как жесткий диск, постоянное запоминающее устройство на основе компакт-диска (CD-ROM), DVD или твердотельный накопитель. Память 1406 содержит инструкции и данные, используемые при помощи процессора 1402. Интерфейс ввода 1414 ввода представляет собой интерфейс на базе сенсорного экрана, мышь, трекбол или другой манипулятор, клавиатуру или их комбинацию и используется для ввода данных в компьютер 1400. В некоторых вариантах осуществления компьютер 1400 может быть сконфигурирован для приема вводных данных (например, команд) из интерфейса ввода 1414 посредством жестов пользователя. Графический адаптер 1412 отображает изображения и другую информацию на дисплее 1418. Сетевой адаптер 1416 соединяет компьютер 1400 с одной или более компьютерными сетями.
[00459] Компьютер 1400 выполнен с возможностью выполнения компьютерных программных модулей для обеспечения функциональности, описанной в данном документе. Используемый в данном документе термин «модуль» относится к компьютерной программной логике, используемой для обеспечения указанной функциональности. Таким образом, модуль может быть реализован в аппаратном обеспечении, программно-аппаратном обеспечении и/или программном обеспечении. В одном варианте осуществления программные модули хранятся в устройстве хранения данных 1408, загружаются в память 1406 и выполняются процессором 1402.
[00460] Типы компьютеров 1400, используемые объектами фиг. 1 может варьироваться в зависимости от варианта осуществления и вычислительной мощности, требуемой объектом. Например, система идентификации презентации 160 может выполняться на одном компьютере 1400 или на нескольких компьютерах 1400, обменивающихся друг с другом через сеть, например, в группе серверов. На компьютерах 1400 могут отсутствовать некоторые из компонентов, описанных выше, таких как графические адаптеры 1412, и дисплеи 1418.
Ссылки
1. Desrichard, A., Snyder, А. & Chan, Т.A. Cancer Neoantigens and Applications for Immunotherapy. Clin. Cancer Res. Off. J. Am. Assoc. Cancer Res. (2015). doi: 10.1158/1078-0432.CCR-14-3175
2. Schumacher, T.N. & Schreiber, R.D. Neoantigens in cancer immunotherapy. Science 348, 69-74 (2015).
3. Gubin, M.M., Artyomov, M.N., Mardis, E.R. & Schreiber, R.D. Tumor neoantigens: building a framework for personalized cancer immunotherapy. J. Clin. Invest. 125, 3413-3421 (2015).
4. Rizvi, N.A. et al. Cancer immunology. Mutational landscape determines sensitivity to PD-1 blockade in non-small cell lung cancer. Science 348, 124-128 (2015).
5. Snyder, A. et al. Genetic basis for clinical response to CTLA-4 blockade in melanoma. N. Engl. J. Med. 371, 2189-2199 (2014).
6. Carreno, В.M. et al. Cancer immunotherapy. A dendritic cell vaccine increases the breadth and diversity of melanoma neoantigen-specific T cells. Science 348, 803-808 (2015).
7. Tran, E. et al. Cancer immunotherapy based on mutation-specific CD4+T cells in a patient with epithelial cancer. Science 344, 641-645 (2014).
8. Hacohen, N. & Wu, C. J.-Y. United States Patent Application: 0110293637 - COMPOSITIONS AND METHODS OF IDENTIFYING TUMOR SPECIFIC NEOANTIGENS. (A1). at <http://appft1.uspto.gov/netacgi/nph-Parser?Sect1=PTO1&Sect2=HITOFF&d=PG01&p=1&u=/netahtml/PTO/srchnum.html&r=1&f=G&l=50&s1=20110293637.PGNR>
9. Lundegaard, C., Hoof, I., Lund, O. & Nielsen, M. State of the art and challenges in sequence based T-cell epitope prediction. Immunome Res. 6 Suppl 2, S3 (2010).
10. Yadav, M. et al. Predicting immunogenic tumour mutations by combining mass spectrometry and exome sequencing. Nature 515, 572-576 (2014).
11. Bassani-Sternberg, M., Pletscher-Frankild, S., Jensen, L. J. & Mann, M. Mass spectrometry of human leukocyte antigen class I peptidomes reveals strong effects of protein abundance and turnover on antigen presentation. Mol. Cell. Proteomics MCP 14, 658-673 (2015).
12. Van Allen, E. M. et al. Genomic correlates of response to CTLA-4 blockade in metastatic melanoma. Science 350, 207-211 (2015).
13. Yoshida, K. & Ogawa, S. Splicing factor mutations and cancer. Wiley Interdiscip.Rev. RNA 5, 445-459 (2014).
14. Cancer Genome Atlas Research Network. Comprehensive molecular profiling of lung adenocarcinoma. Nature 511, 543-550 (2014).
15. Rajasagi, M. et al. Systematic identification of personal tumor-specific neoantigens in chronic lymphocytic leukemia. Blood 124, 453-462 (2014).
16. Downing, S.R. et al. United States Patent Application: 0120208706 - OPTIMIZATION OF MULTIGENE ANALYSIS OF TUMOR SAMPLES. (A1). at <http://appft1.uspto.gov/netacgi/nph-Parser?Sect1=PTO1&Sect2=HITOFF&d=PG01&p=1&u=/netahtml/PTO/srchnum.html&r=1&f=G&1=50&s1=20120208706.PGNR.>
17. Target Capture for NextGen Sequencing - IDT. at <http://www.idtdna.com/pages/products/nextgen/target-capture>
18. Shukla, S.A. et al. Comprehensive analysis of cancer-associated somatic mutations in class I HLA genes. Nat. Biotechnol. 33, 1152-1158 (2015).
19. Cieslik, M. et al. The use of exome capture RNA-seq for highly degraded RNA with application to clinical cancer sequencing. Genome Res. 25, 1372-1381 (2015).
20. Bodini, M. et al. The hidden genomic landscape of acute myeloid leukemia: subclonal structure revealed by undetected mutations. Blood 125, 600-605 (2015).
21. Saunders, С.T. et al. Strelka: accurate somatic small-variant calling from sequenced tumor-normal sample pairs. Bioinforma. Oxf. Engl. 28, 1811-1817 (2012).
22. Cibulskis, K. et al. Sensitive detection of somatic point mutations in impure and heterogeneous cancer samples. Nat. Biotechnol. 31, 213-219 (2013).
23. Wilkerson, M.D. et al. Integrated RNA and DNA sequencing improves mutation detection in low purity tumors. Nucleic Acids Res. 42, e107 (2014).
24. Mose, L.E., Wilkerson, M.D., Hayes, D.N., Perou, С.M. & Parker, J.S. ABRA: improved coding indel detection via assembly-based realignment. Bioinforma. Oxf. Engl. 30, 2813-2815 (2014).
25. Ye, K., Schulz, M. H., Long, Q., Apweiler, R. & Ning, Z. Pindel: a pattern growth approach to detect break points of large deletions and medium sized insertions from paired-end short reads. Bioinforma. Oxf Engl. 25, 2865-2871 (2009).
26. Lam, H. Y. K. et al. Nucleotide-resolution analysis of structural variants using BreakSeq and a breakpoint library. Nat. Biotechnol. 28, 47-55 (2010).
27. Frampton, G.M. et al. Development and validation of a clinical cancer genomic profiling test based on massively parallel DNA sequencing. Nat. Biotechnol. 31, 1023-1031 (2013).
28. Boegel, S. et al. HLA typing from RNA-Seq sequence reads. Genome Med. 4, 102 (2012).
29. Liu, C. et al. ATHLATES: accurate typing of human leukocyte antigen through exome sequencing. Nucleic Acids Res. 41, e142 (2013).
30. Mayor, N. P. et al. HLA Typing for the Next Generation. PloS One 10, e0127153 (2015).
31. Roy, С.K., Olson, S., Graveley, B.R., Zamore, P.D. & Moore, M. J. Assessing long-distance RNA sequence connectivity via RNA-templated DNA-DNA ligation. eLife 4, (2015).
32. Song, L. & Florea, L. CLASS: constrained transcript assembly of RNA-seq reads. BMC Bioinformatics 14 Suppl 5, S14 (2013).
33. Maretty, L., Sibbesen, J.A. & Krogh, A. Bayesian transcriptome assembly. Genome Biol. 15, 501 (2014).
34. Pertea, M. et al. StringTie enables improved reconstruction of a transcriptome from RNA-seq reads. Nat. Biotechnol. 33, 290-295 (2015).
35. Roberts, A., Pimentel, H., Trapnell, C. & Pachter, L. Identification of novel transcripts in annotated genomes using RNA-Seq. Bioinforma. Oxf. Engl. (2011). doi: 10.1093/bioinformatics/btr355
36. Vitting-Seerup, K., Porse, В.Т., Sandelin, A. & Waage, J. spliceR: an R package for classification of alternative splicing and prediction of coding potential from RNA-seq data. BMC Bioinformatics 15, 81 (2014).
37. Rivas, M.A. et al. Human genomics. Effect of predicted protein-truncating genetic variants on the human transcriptome. Science 348, 666-669 (2015).
38. Skelly, D.A., Johansson, M., Madeoy, J., Wakefield, J. & Akey, J.M. A powerful and flexible statistical framework for testing hypotheses of allele-specific gene expression from RNA-seq data. Genome Res. 21, 1728-1737 (2011).
39. Anders, S., Pyl, P.T. & Huber, W. HTSeq--a Python framework to work with high-throughput sequencing data. Bioinforma. Oxf. Engl. 31, 166-169 (2015).
40. Furney, S.J. et al. SF3B1 mutations are associated with alternative splicing in uveal melanoma. Cancer Discov. (2013). doi:10.1158/2159-8290.CD-13-0330
41. Zhou, Q. et al. A chemical genetics approach for the functional assessment of novel cancer genes. Cancer Res. (2015). doi:10.1158/0008-5472.CAN-14-2930
42. Maguire, S.L. et al. SF3B1 mutations constitute a novel therapeutic target in breast cancer. J. Pathol. 235, 571-580 (2015).
43. Carithers, L.J. et al. A Novel Approach to High-Quality Postmortem Tissue Procurement: The GTEx Project. Biopreservation Biobanking 13, 311-319 (2015).
44. Xu, G. et al. RNA CoMPASS: a dual approach for pathogen and host transcriptome analysis of RNA-seq datasets. PloS One 9, e89445 (2014).
45. Andreatta, M. & Nielsen, M. Gapped sequence alignment using artificial neural networks: application to the MHC class I system. Bioinforma. Oxf. Engl. (2015). doi: 10.1093-bioinformatics/btv639
46. Jorgensen, K.W., Rasmussen, M., Buus, S. & Nielsen, M. NetMHCstab - predicting stability of peptide-MHC-I complexes; impacts for cytotoxic T lymphocyte epitope discovery. Immunology 141, 18-26 (2014).
47. Larsen, M.V. et al. An integrative approach to CTL epitope prediction: a combined algorithm integrating MHC class I binding, TAP transport efficiency, and proteasomal cleavage predictions. Eur. Immunol. 35, 2295-2303 (2005).
48. Nielsen, M., Lundegaard, C., Lund, O. & Kesmir, C. The role of the proteasome in generating cytotoxic T-cell epitopes: insights obtained from improved predictions of proteasomal cleavage. Immunogenetics 57, 33-41 (2005).
49. Boisvert, F.-M. et al. A Quantitative Spatial Proteomics Analysis of Proteome Turnover in Human Cells. Mol. Cell. Proteomics 11, M111.011429-M111.011429 (2012).
50. Duan, F. et al. Genomic and bioinformatic profiling of mutational neoepitopes reveals new rules to predict anticancer immunogenicity. J. Exp. Med. 211, 2231-2248 (2014).
51.  Immunobiology: 9780815345312: Medicine & Health Science Books @ Amazon.com, at <http://www.amazon.com/Janeways-Immunobiology-Kenneth-Murphy/dp/0815345313>
Immunobiology: 9780815345312: Medicine & Health Science Books @ Amazon.com, at <http://www.amazon.com/Janeways-Immunobiology-Kenneth-Murphy/dp/0815345313>
52. Calis, J. J. A. et al. Properties of MHC Class I Presented Peptides That Enhance Immunogenicity. PLoS Comput. Biol. 9, el003266 (2013).
53. Zhang, J. et al. Intratumor heterogeneity in localized lung adenocarcinomas delineated by multiregion sequencing. Science 346, 256-259 (2014)
54. Walter, M. J. et al. Clonal architecture of secondary acute myeloid leukemia. N. Engl. J. Med. 366, 1090-1098 (2012).
55. Hunt DF, Henderson RA, Shabanowitz J, Sakaguchi K, Michel H, Sevilir N, Cox AL, Appella E, Engelhard VH. Characterization of peptides bound to the class IMHC molecule HLA-A2.1 by mass spectrometry. Science 1992. 255: 1261-1263.
56. Zarling AL, Polefrone JM, Evans AM, Mikesh LM, Shabanowitz J, Lewis ST, Engelhard VH, Hunt DF. Identification of class I МНС-associated phosphopeptides as targets for cancer immunotherapy. Proc Natl Acad Sci USA. 2006 Oct 3; 103(40):14889-94.
57. Bassani-Sternberg M, Pletscher-Frankild S, Jensen LJ, Mann M. Mass spectrometry of human leukocyte antigen class I peptidomes reveals strong effects of protein abundance and turnover on antigen presentation. Mol Cell Proteomics. 2015 Mar; 14(3):658-73. doi: 10.1074/mcp.M114.042812.
58. Abelin JG, Trantham PD, Penny SA, Patterson AM, Ward ST, Hildebrand WH, Cobbold M, Bai DL, Shabanowitz J, Hunt DF. Complementary IMAC enrichment methods for HLA-associated phosphopeptide identification by mass spectrometry. Nat Protoc. 2015 Sep; 10(9): 1308-18. doi: 10.1038/nprot.2015.086. Epub 2015 Aug 6
59. Barnstable CJ, Bodmer WF, Brown G, Galfre G, Milstein C, Williams AF, Ziegler A. Production of monoclonal antibodies to group A erythrocytes, HLA and other human cell surface antigens-new tools for genetic analysis. Cell. 1978 May;14(l):9-20.
60. Goldman JM, Hibbin J, Kearney L, Orchard K,  KH. HLA-DR monoclonal antibodies inhibit the proliferation of normal and chronic granulocytic leukaemia myeloid progenitor cells. Br J Haematol. 1982 Nov; 52(3):411-20.
KH. HLA-DR monoclonal antibodies inhibit the proliferation of normal and chronic granulocytic leukaemia myeloid progenitor cells. Br J Haematol. 1982 Nov; 52(3):411-20.
61. Eng JK, Jahan ТА, Hoopmann MR. Comet: an open-source MS/MS sequence database search tool. Proteomics. 2013 Jan; 13(l):22-4. doi: 10.1002/pmic.201200439. Epub 2012 Dec 4.
62. Eng JK, Hoopmann MR, Jahan ТА, Egertson JD, Noble WS, MacCoss MJ. A deeper look into Comet-implementation and features. J Am Soc Mass Spectrom. 2015 Nov; 26(11):1865-74.doi: 10.1007/s13361-015-1179-x. Epub 2015 Jun 27.
63. Lukas  , Jesse Canterbury, Jason Weston, William Stafford Noble and Michael J. MacCoss. Semi-supervised learning for peptide identification from shotgun proteomics datasets. Nature Methods 4:923-925, November 2007
, Jesse Canterbury, Jason Weston, William Stafford Noble and Michael J. MacCoss. Semi-supervised learning for peptide identification from shotgun proteomics datasets. Nature Methods 4:923-925, November 2007
64. Lukas  , John D. Storey, Michael J. MacCoss and William Stafford Noble. Assigning confidence measures to peptides identified by tandem mass spectrometry. Journal of Proteome Research, 7(l):29-34, January 2008
, John D. Storey, Michael J. MacCoss and William Stafford Noble. Assigning confidence measures to peptides identified by tandem mass spectrometry. Journal of Proteome Research, 7(l):29-34, January 2008
65. Lukas  , John D. Storey and William Stafford Noble. Nonparametric estimation of posterior error probabilities associated with peptides identified by tandem mass spectrometry. Bioinformatics, 24(16):i42-i48, August 2008
, John D. Storey and William Stafford Noble. Nonparametric estimation of posterior error probabilities associated with peptides identified by tandem mass spectrometry. Bioinformatics, 24(16):i42-i48, August 2008
66. Bo Li and Colin N. Dewey. RSEM: accurate transcript quantification from RNA-Seq data with or without a referenfe genome. BMC Bioinformatics, 12:323, August 2011
67. Hillary Pearson, Tariq Daouda, Diana Paola Granados, Chantal Durette, Eric Bonneil, Mathieu Courcelles, Anja Rodenbrock, Jean-Philippe Laverdure, Caroline  , Sylvie Mader, Sebastien Lemieux, Pierre Thibault, and Claude Perreault. MHC class I-associated peptides derive from selective regions of the human genome. The Journal of Clinical Investigation, 2016,
, Sylvie Mader, Sebastien Lemieux, Pierre Thibault, and Claude Perreault. MHC class I-associated peptides derive from selective regions of the human genome. The Journal of Clinical Investigation, 2016,
68. Juliane Liepe, Fabio Marino, John Sidney, Anita Jeko, Daniel E. Bunting, Alessandro Sette, Peter M. Kloetzel, Michael P. H. Stumpf, Albert J. R. Heck, Michele Mishto. A large fraction of HLA class I ligands are proteasome-generated spliced peptides. Science, 21, October 2016.





























| название | год | авторы | номер документа |
|---|---|---|---|
| НЕОАНТИГЕННЫЕ ВЕКТОРЫ НА ОСНОВЕ АЛЬФАВИРУСА | 2018 |
|
RU2803566C2 |
| СПОСОБ И СИСТЕМЫ ДЛЯ ПРОГНОЗИРОВАНИЯ СПЕЦИФИЧЕСКИХ ДЛЯ HLA КЛАССА II ЭПИТОПОВ И ОХАРАКТЕРИЗАЦИИ CD4+ T-КЛЕТОК | 2019 |
|
RU2826261C2 |
| Выбор раковых мутаций для создания персонализированной противораковой вакцины | 2019 |
|
RU2809620C2 |
| ПРЕДСКАЗАНИЕ ИММУНОГЕННОСТИ Т-КЛЕТОЧНЫХ ЭПИТОПОВ | 2014 |
|
RU2724370C2 |
| СПОСОБ ЛЕЧЕНИЯ РАКА | 2016 |
|
RU2770447C2 |
| СПОСОБЫ ПРОГНОЗИРОВАНИЯ ПРИМЕНИМОСТИ СПЕЦИФИЧНЫХ ДЛЯ ЗАБОЛЕВАНИЯ АМИНОКИСЛОТНЫХ МОДИФИКАЦИЙ ДЛЯ ИММУНОТЕРАПИИ | 2018 |
|
RU2799341C2 |
| НЕОАНТИГЕННЫЕ КОМПОЗИЦИИ И ИХ ИСПОЛЬЗОВАНИЕ | 2020 |
|
RU2832574C2 |
| ПОЛУЧЕНИЕ АНТИГЕНСПЕЦИФИЧЕСКИХ T-КЛЕТОК | 2017 |
|
RU2745319C2 |
| МЕТОД | 2016 |
|
RU2739036C2 |
| СПОСОБЫ ПРОГНОЗИРОВАНИЯ ПРИМЕНИМОСТИ БЕЛКОВ ИЛИ БЕЛКОВЫХ ФРАГМЕНТОВ ДЛЯ ИММУНОТЕРАПИИ | 2017 |
|
RU2782336C2 |
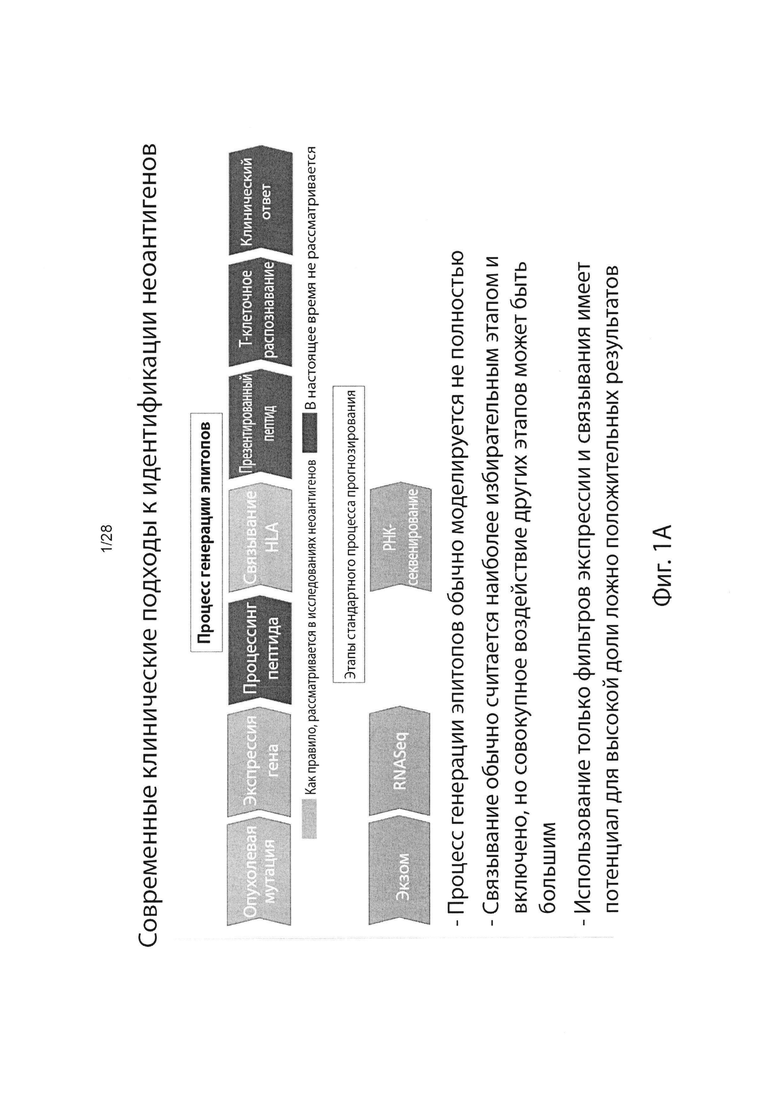

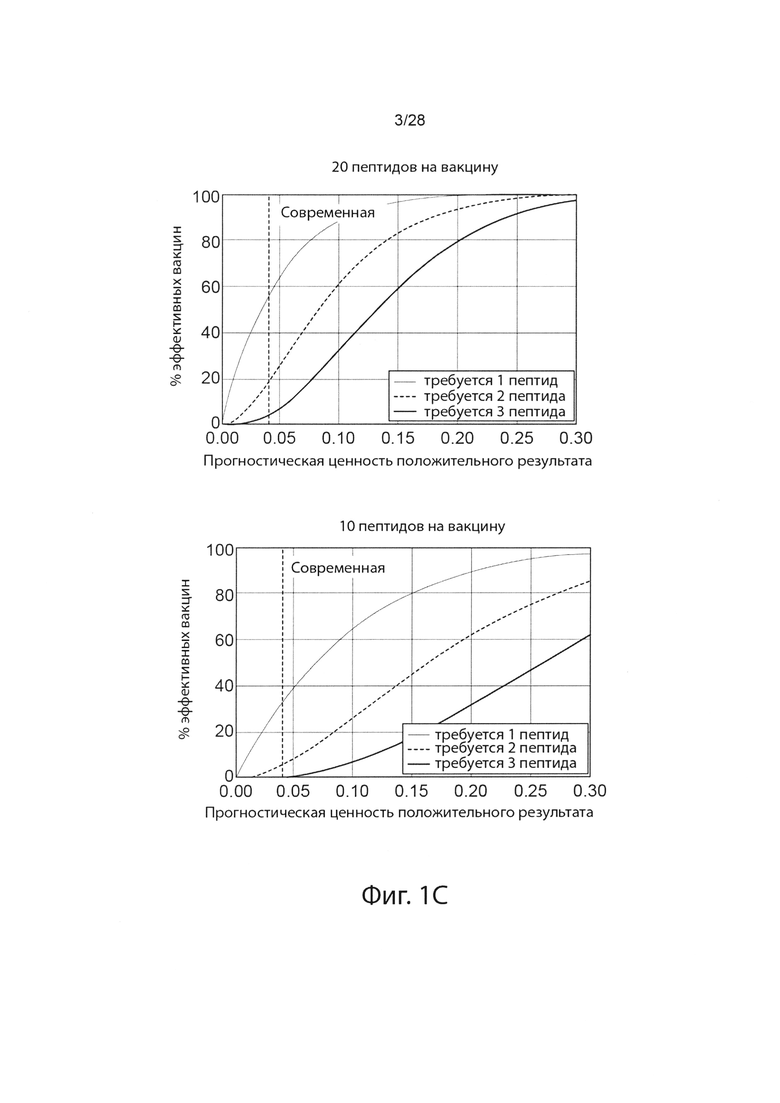
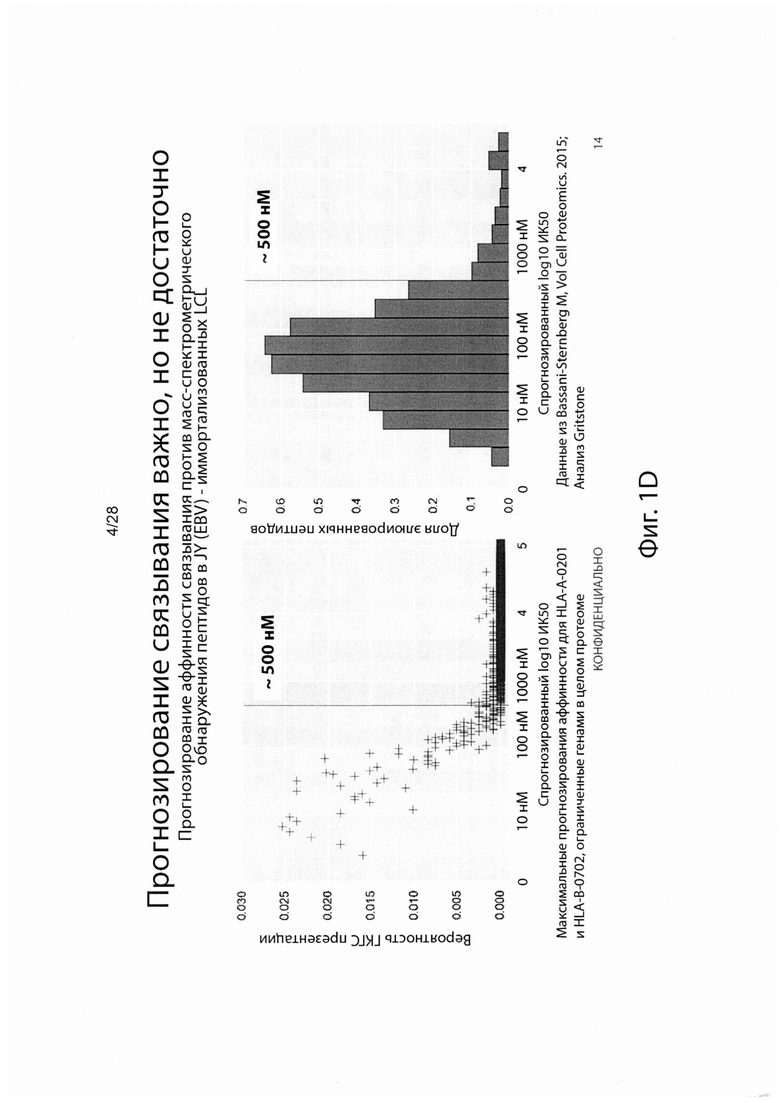

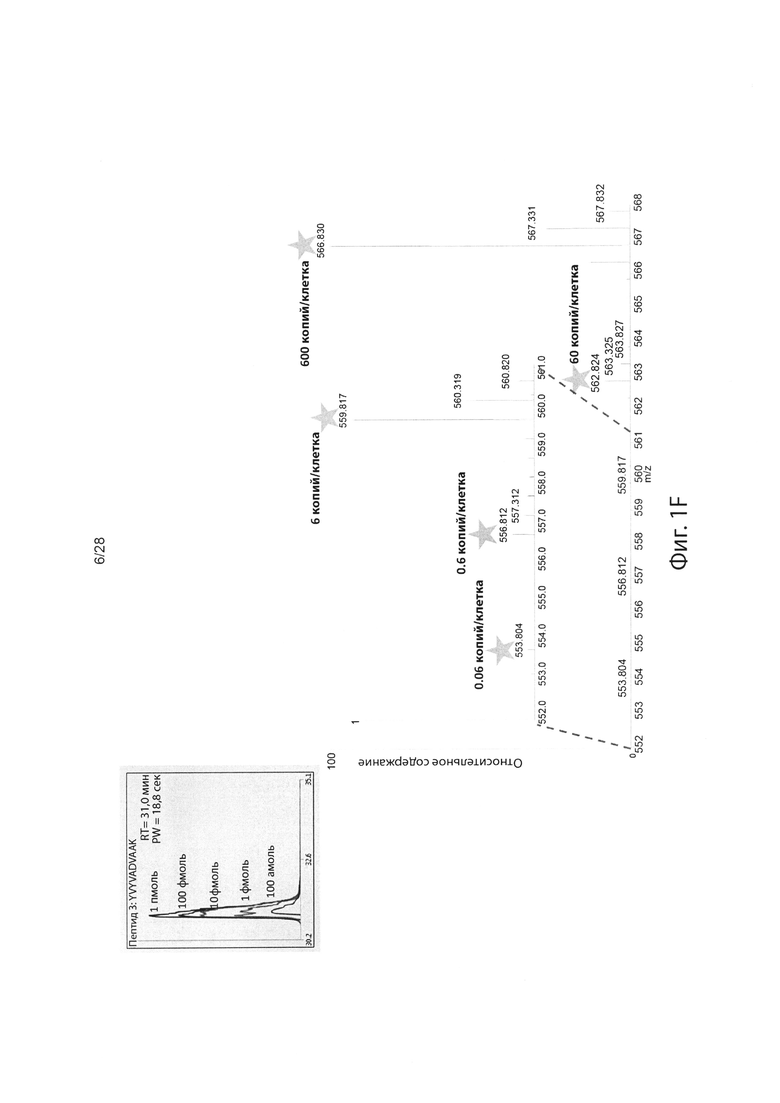

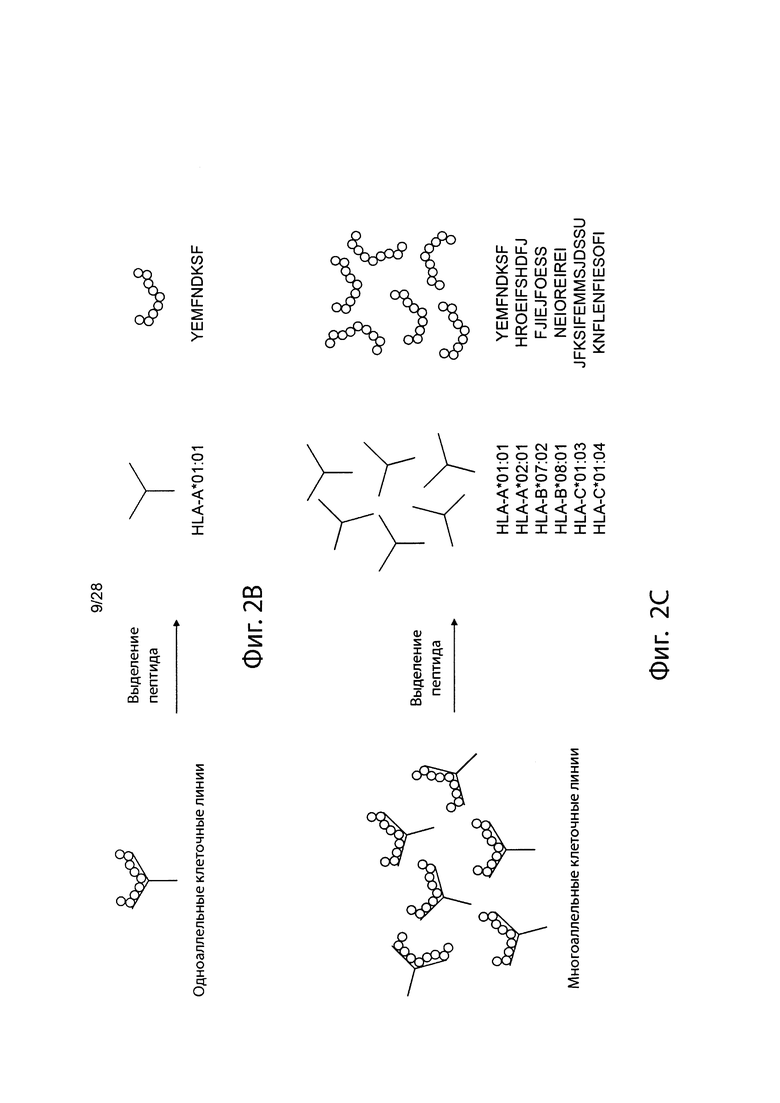
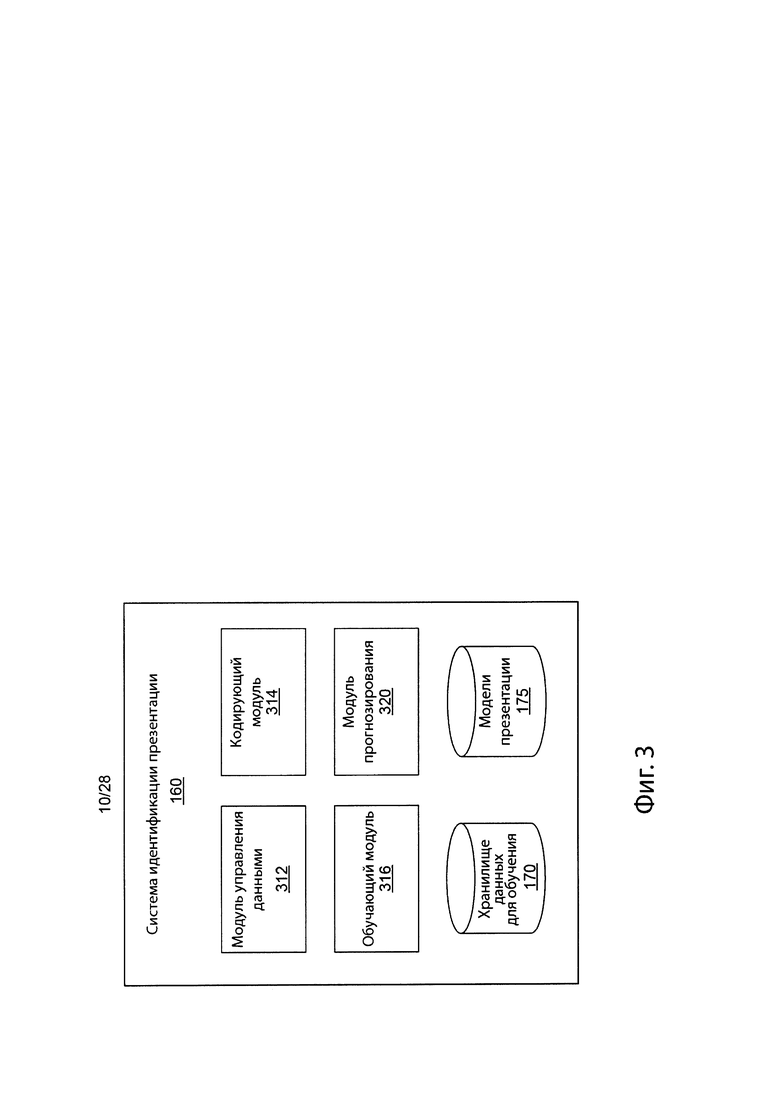
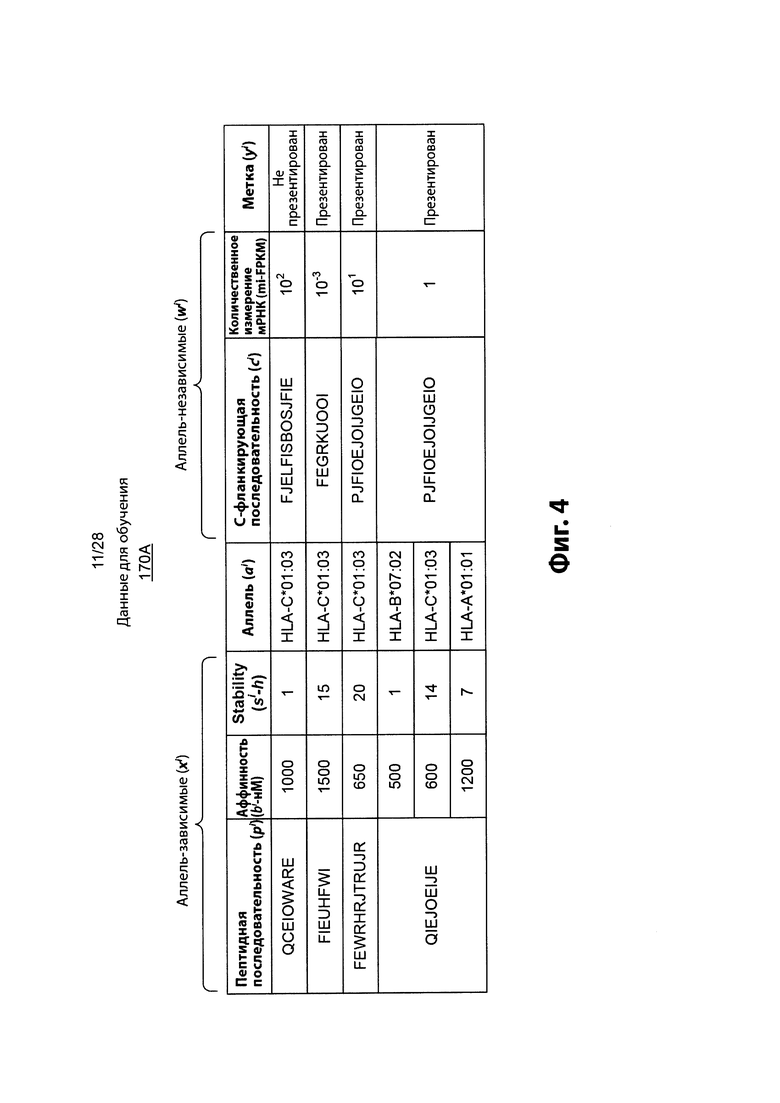
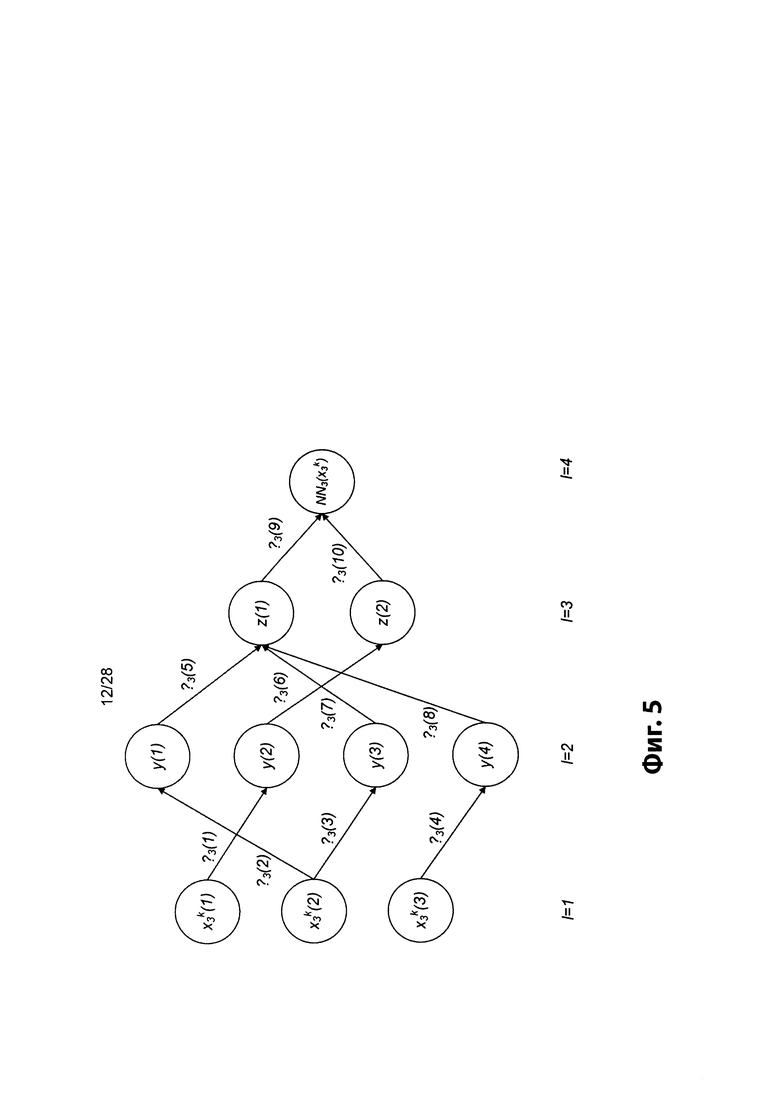
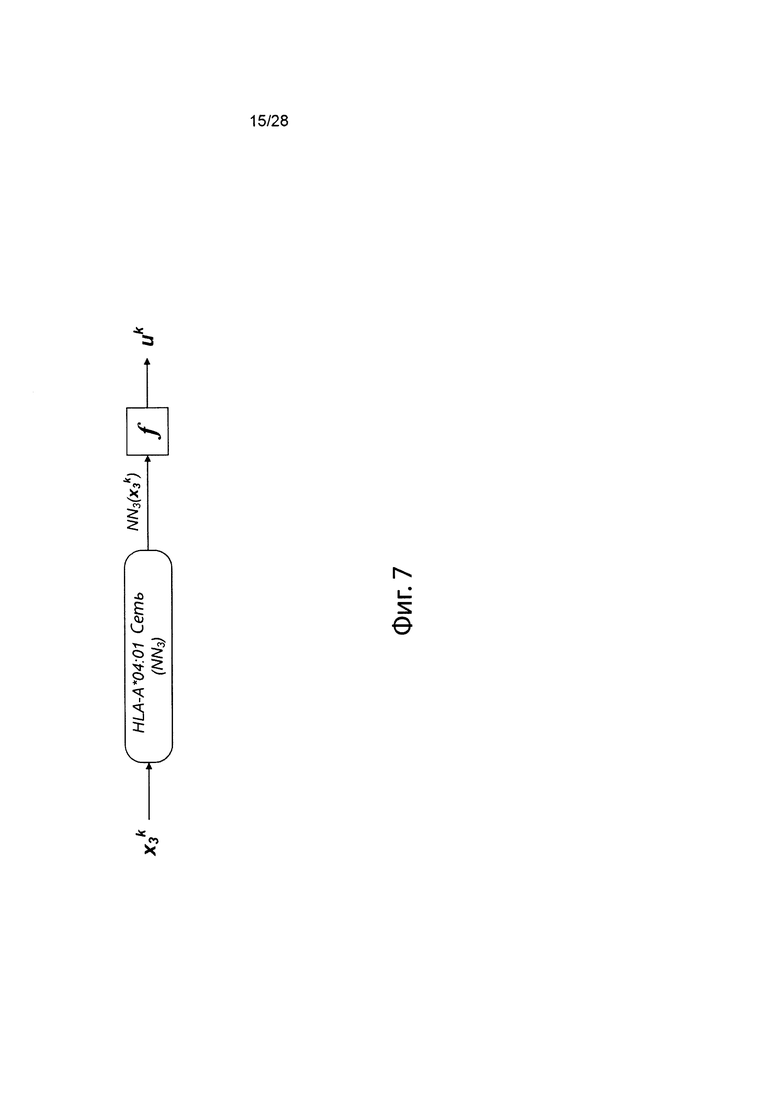
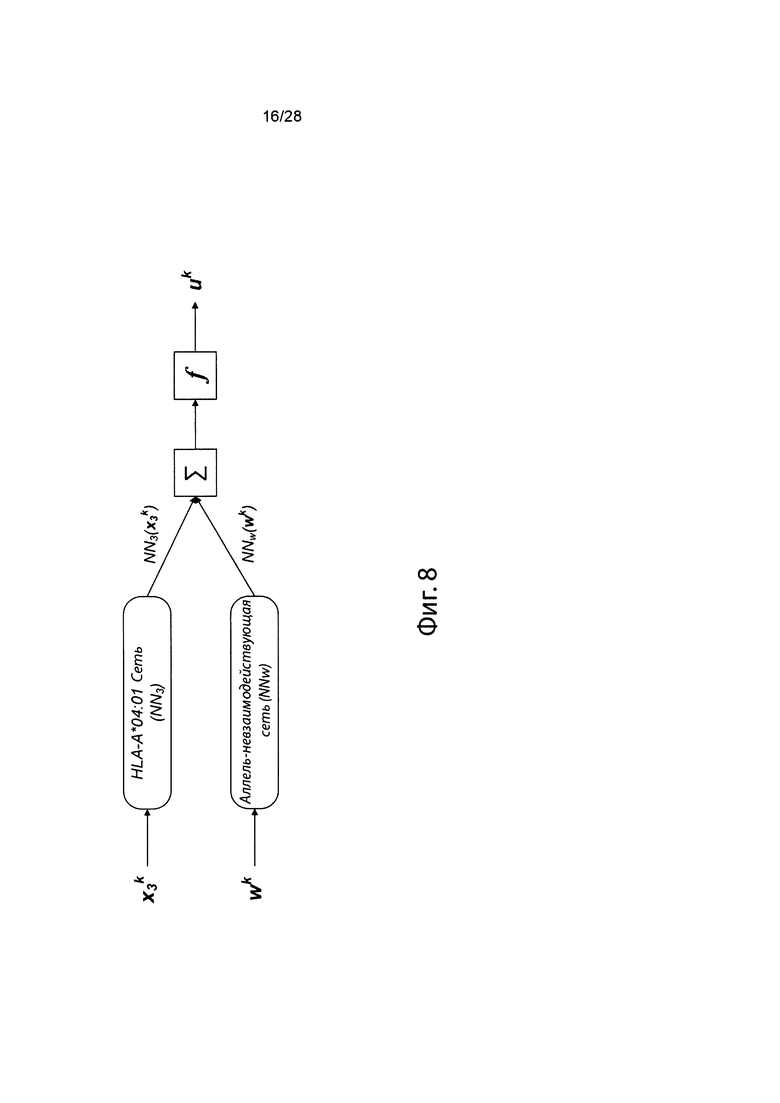
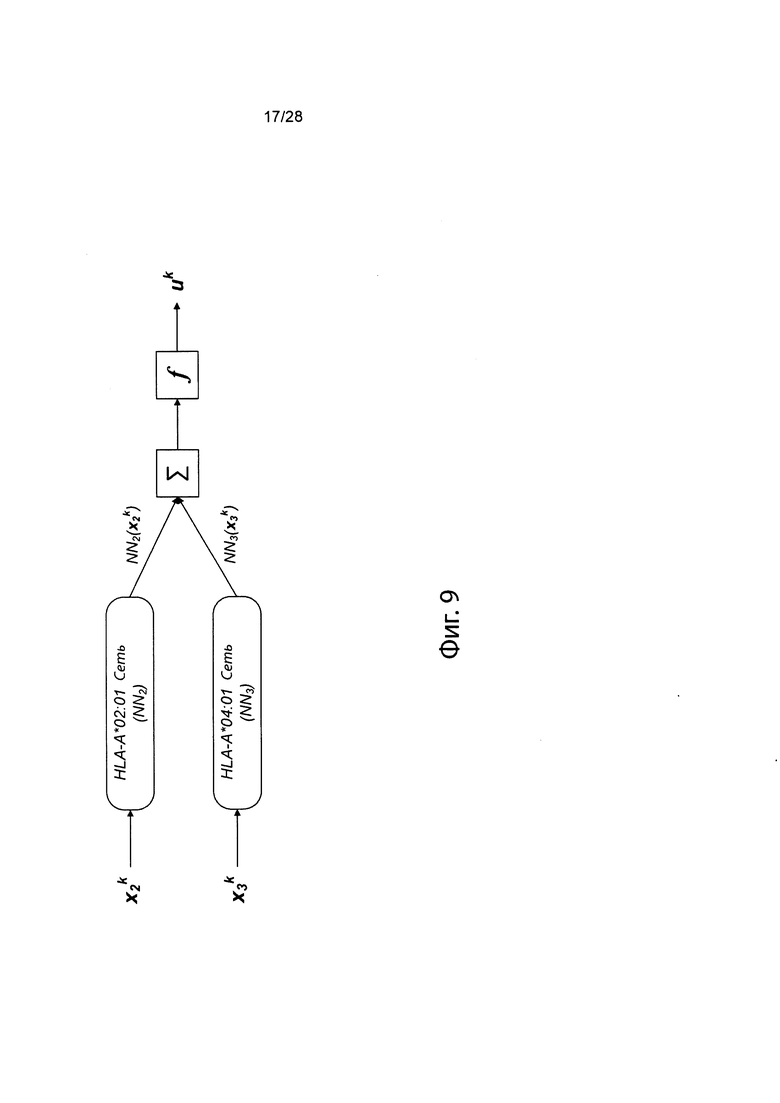
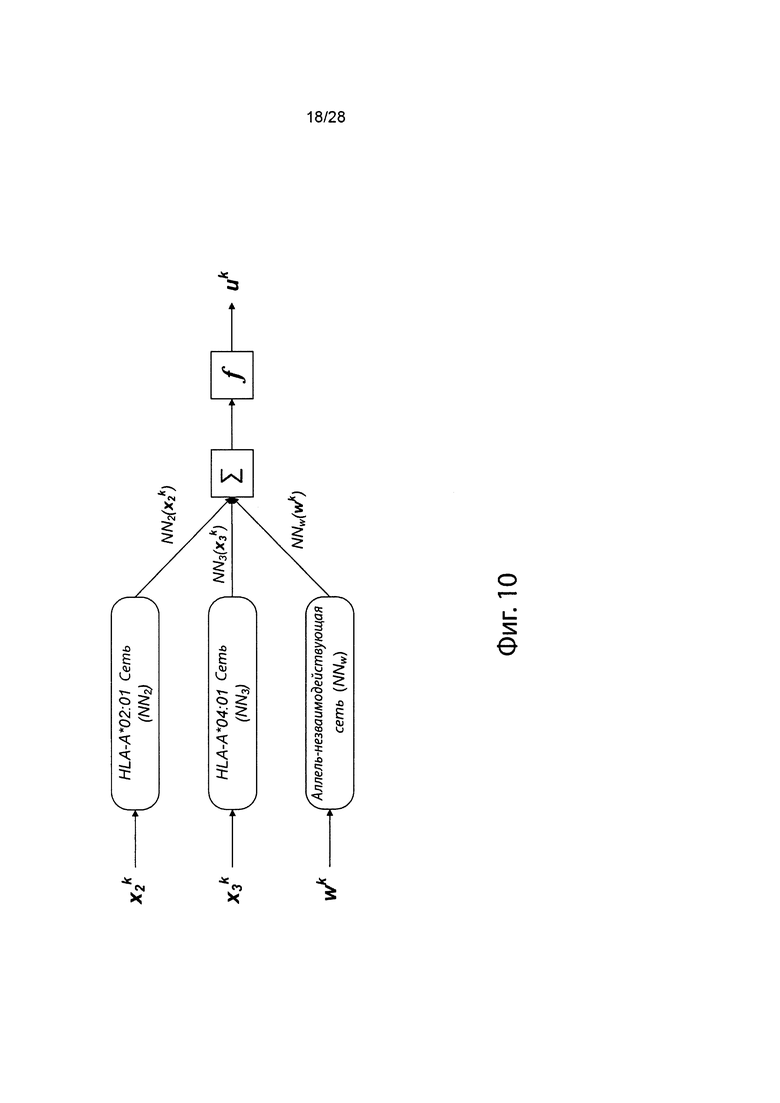

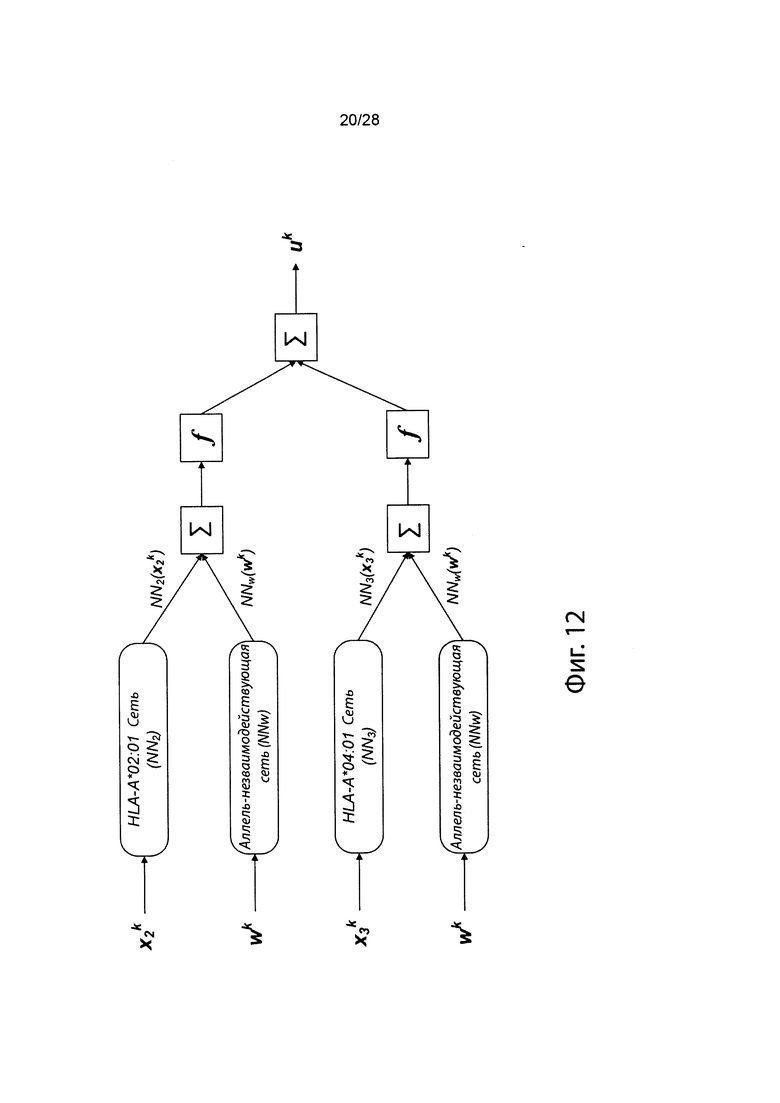
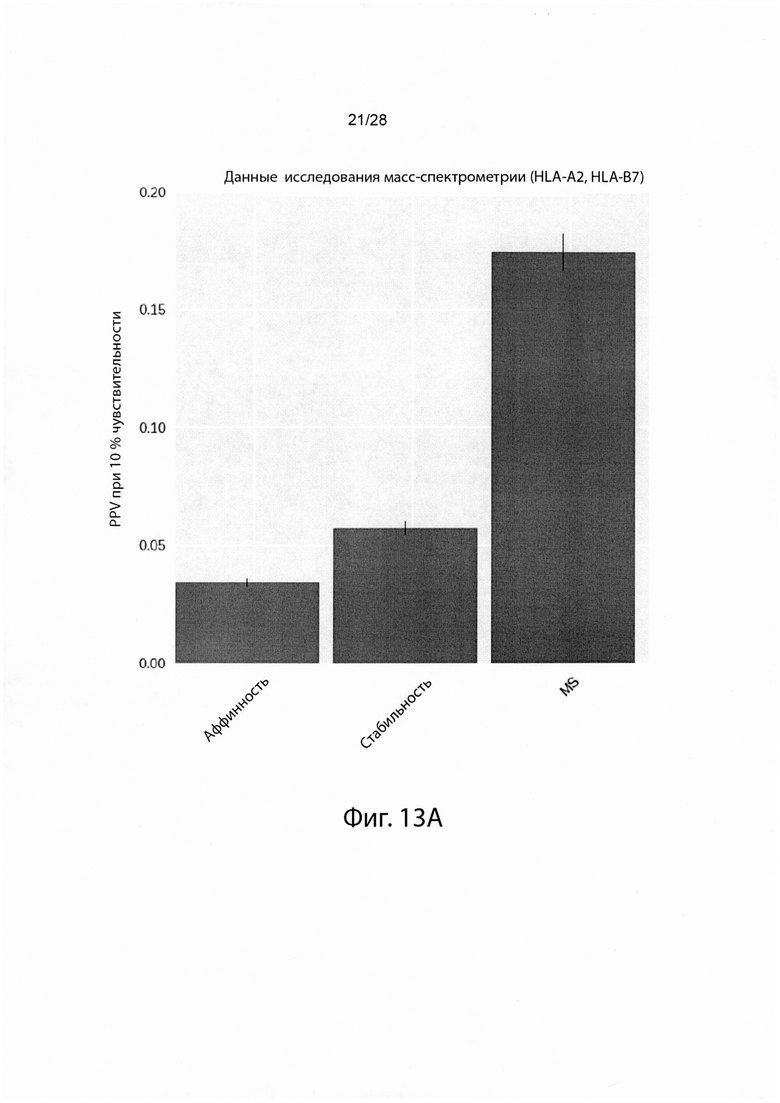
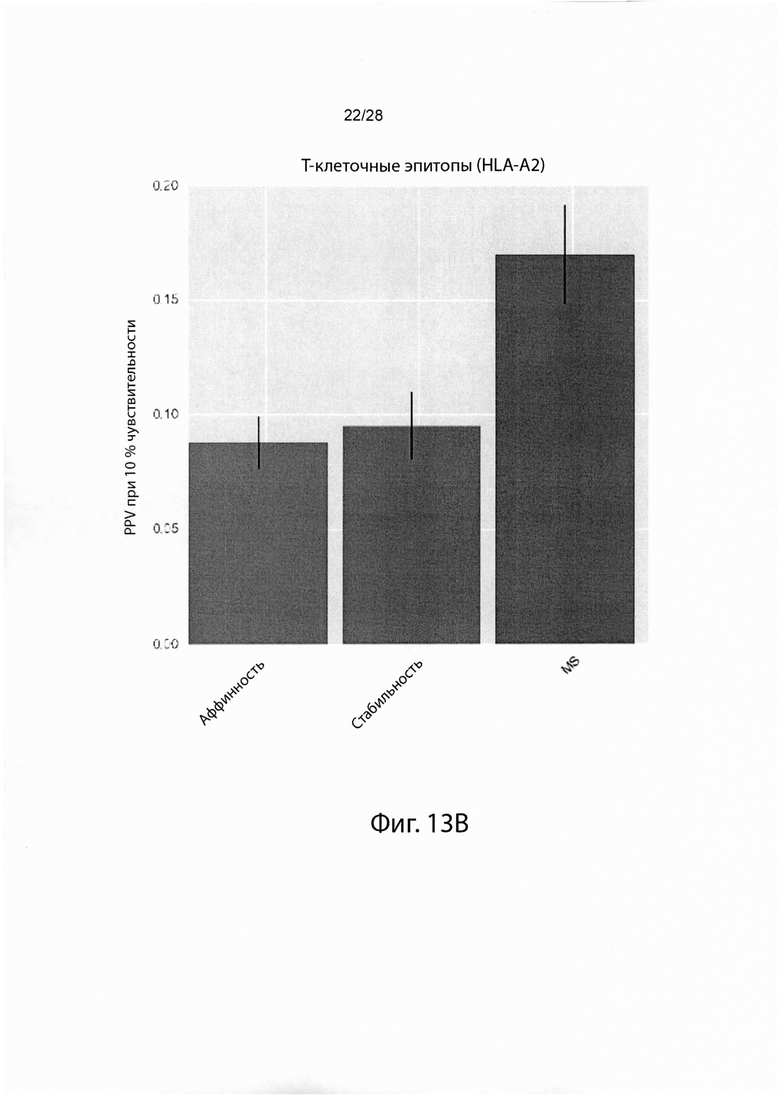


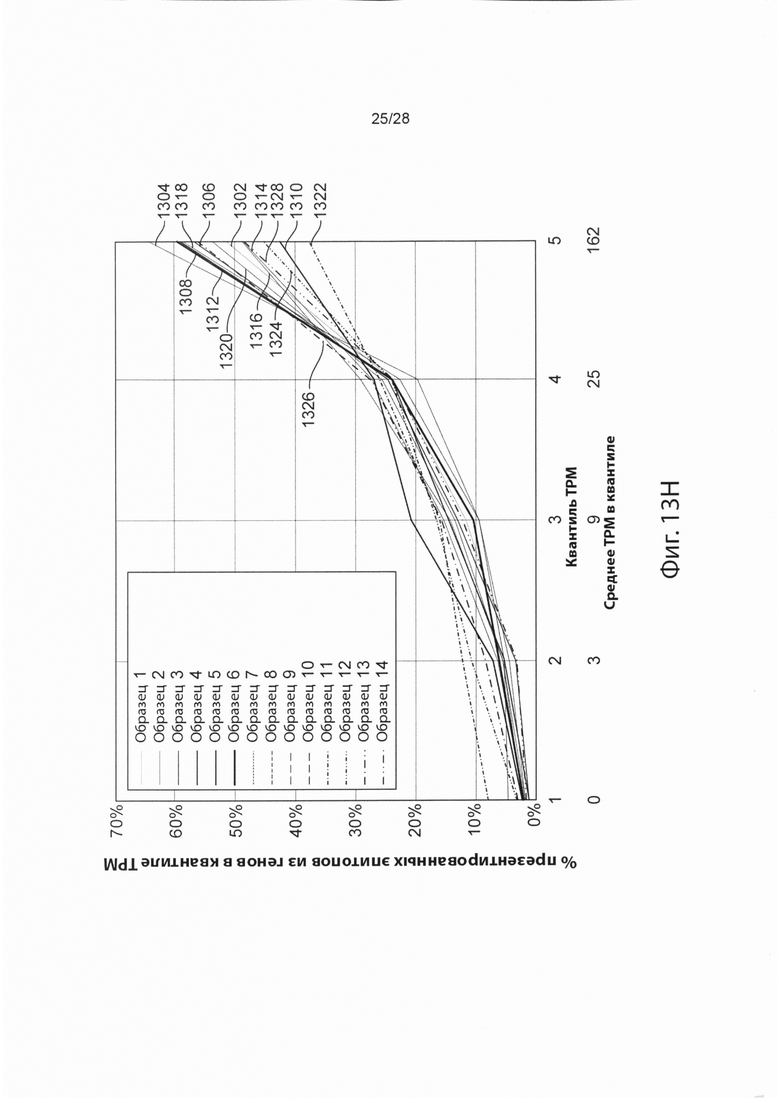
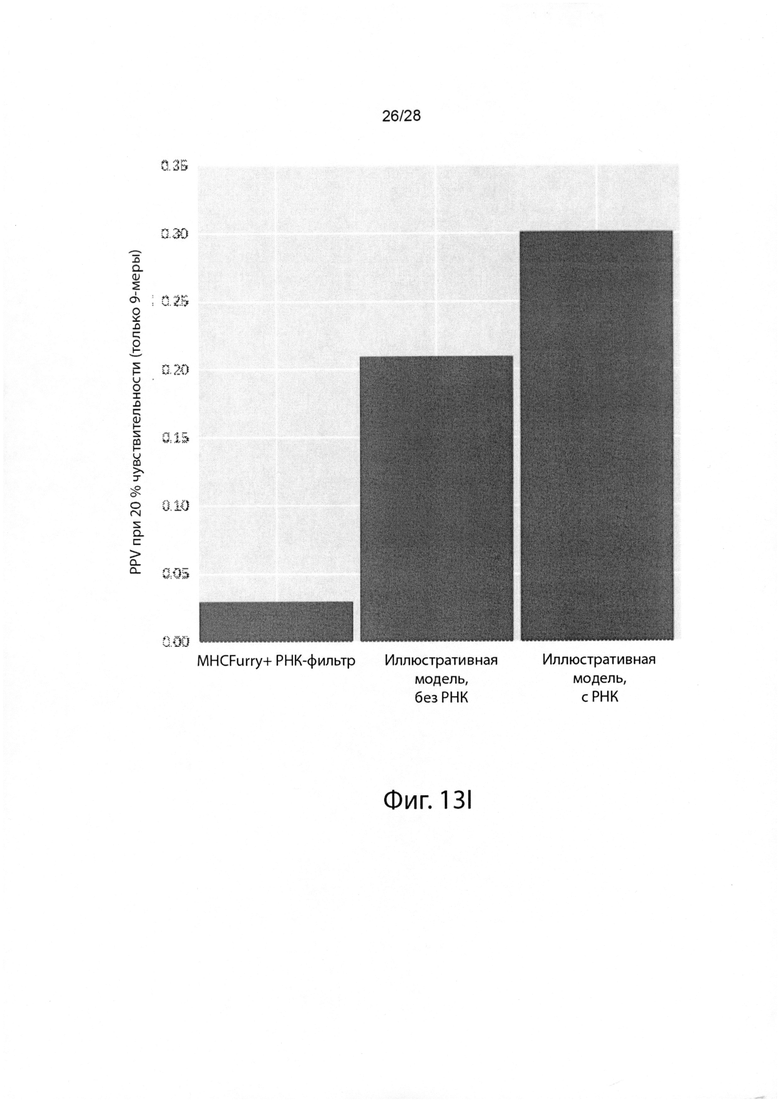
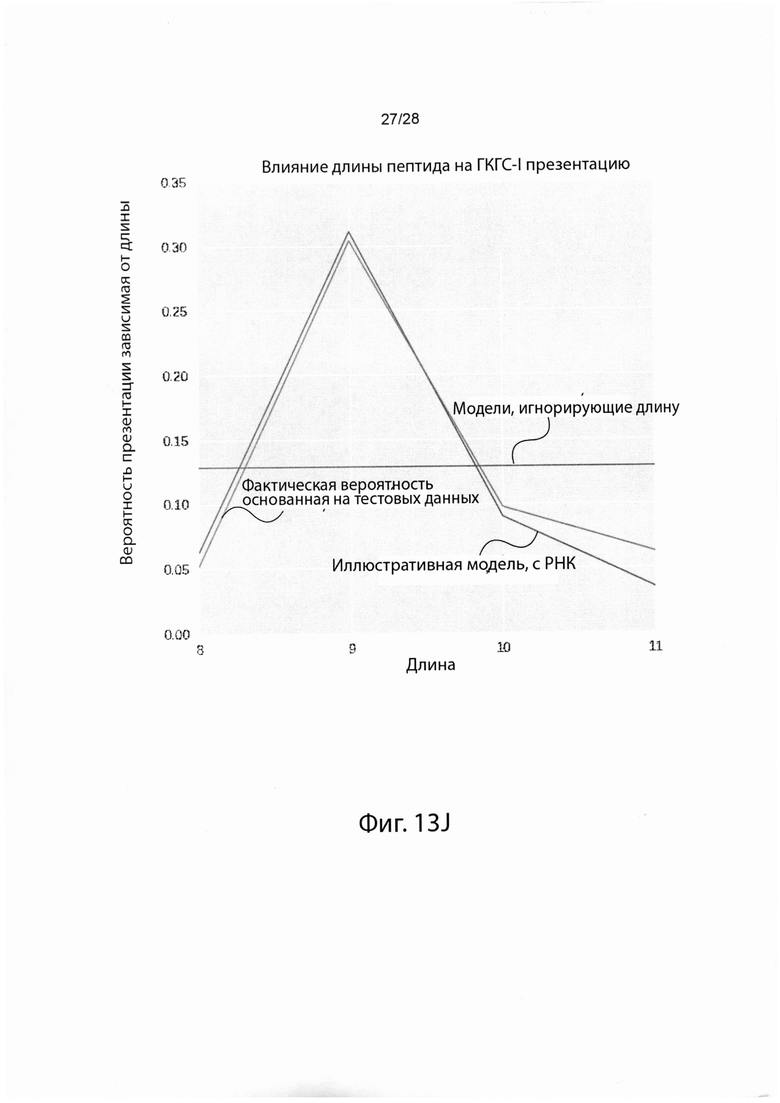

Изобретение относится к области биотехнологии, конкретно к способу идентификации одного или более неоантигенов из опухолевой клетки, и может быть использовано в медицине. Предложенный способ идентификации неоантигенов, включающий моделирование презентации неоантигенов, может быть использован для получения противораковых вакцин. 6 н. и 72 з.п. ф-лы, 14 ил., 1 табл., 6 пр.
1. Способ идентификации одного или более неоантигенов из опухолевой клетки субъекта, которые имеют вероятность быть презентированными на поверхности опухолевых клеток, включающий этапы:
получения по меньшей мере одного из данных нуклеотидного секвенирования экзома, транскриптома или всего генома опухоли из опухолевой клетки субъекта, причем данные нуклеотидного секвенирования опухоли используются для получения данных, представляющих пептидные последовательности каждой из совокупности неоантигенов, и при этом пептидная последовательность каждого неоантигена содержит по меньшей мере одно изменение, которое отличает его от соответствующей исходной последовательности пептида дикого типа;
ввода пептидной последовательности каждого неоантигена в одну или более моделей презентации для создания совокупности числовых значений вероятностей того, что каждый из неоантигенов презентирован одним или более аллелями ГКГС на поверхности опухолевой клетки опухолевой клетки субъекта, при этом совокупность числовых значений вероятностей была идентифицирована по меньшей мере на основе полученных данных масс-спектрометрии; и
выбора подсовокупности из совокупности неоантигенов на основе совокупности числовых значений вероятностей для создания совокупности выбранных неоантигенов.
2. Способ по п. 1, отличающийся тем, что размер совокупности выбранных неоантигенов составляет 20.
3. Способ по любому из пп. 1, 2, отличающийся тем, что модель презентации представляет зависимость между:
присутствием пары определенного одного из аллелей ГКГС и определенной аминокислоты в определенном положении пептидной последовательности; и
вероятностью презентации на поверхности опухолевой клетки, одним определенным ГКГС аллелем указанной пары, такой пептидной последовательности, содержащей определенную аминокислоту в определенном положении.
4. Способ по любому из пп. 1-3, отличающийся тем, что ввод пептидной последовательности включает:
применение одной или более моделей презентации к пептидной последовательности соответствующего неоантигена для генерации оценки зависимости для каждого из одного или более аллелей ГКГС, указывающих, будет ли аллель ГКГС презентировать соответствующий неоантиген, на основании по меньшей мере положения аминокислот пептидной последовательности соответствующего неоантигена.
5. Способ по п. 4, дополнительно включающий:
преобразование оценок зависимостей для генерации соответствующей вероятности на аллель для каждого аллеля ГКГС, указывающего вероятность того, что соответствующий аллель ГКГС будет презентировать соответствующий неоантиген; и
комбинирование вероятностей на аллель для генерации числового значения вероятности.
6. Способ по п. 5, отличающийся тем, что преобразование оценок зависимостей моделирует презентацию пептидной последовательности соответствующего неоантигена как взаимоисключающих.
7. Способ по любому из пп. 4-6, дополнительно включающий:
преобразование комбинации оценок зависимостей для генерации числового значения вероятности.
8. Способ по п. 7, отличающийся тем, что преобразование комбинации оценок зависимостей моделирует презентацию пептидной последовательности соответствующего неоантигена как интерферирующего между аллелями ГКГС.
9. Способ по любому из пп. 4-8, отличающийся тем, что совокупность числовых значений вероятностей дополнительно идентифицируется по меньшей мере аллель-невзаимодействующим признаком и дополнительно включающий:
применение аллель-невзаимодействующей модели из одной или более моделей презентации к аллель-невзаимодействующим признакам для генерации оценки зависимости для аллель-невзаимодействующих признаков, указывающих, будет ли презентирована пептидная последовательность соответствующего неоантигена на основе аллель-невзаимодействующих признаков.
10. Способ по п. 9, дополнительно включающий:
комбинирование оценки зависимости для каждого аллеля ГКГС в одном или более аллелях ГКГС с оценкой зависимости для аллель-невзаимодействующего признака;
преобразование комбинированных оценок зависимостей для каждого аллеля ГКГС для генерации соответствующей вероятности на аллель для каждого аллеля ГКГС, указывающего вероятность того, что соответствующий аллель ГКГС будет презентировать соответствующий неоантиген; и
комбинирование вероятностей на аллель для генерации числового значения вероятности.
11. Способ по любому из пп. 9, 10, дополнительно включающий:
преобразование комбинации оценок зависимостей для каждого из аллелей ГКГС и оценки зависимости для аллель-невзаимодействующих признаков для генерации числового значения вероятности.
12. Способ по любому из пп. 1-11, отличающийся тем, что совокупность числовых параметров для модели презентации обучена на основе совокупности данных для обучения, включающего по меньшей мере совокупность пептидных последовательностей для обучения, идентифицированных как присутствующие во множестве образцов, и один или более аллелей ГКГС, связанных с каждой пептидной последовательностью для обучения, при этом пептидные последовательности для обучения идентифицируют с помощью масс-спектрометрии на выделенных пептидах, элюированных из аллелей ГКГС, полученных из множества образцов.
13. Способ по п. 12, отличающийся тем, что совокупность данных для обучения дополнительно включает данные об уровнях экспрессии мРНК опухолевой клетки.
14. Способ по любому из пп. 12, 13, отличающийся тем, что образцы содержат линии клеток, модифицированные для экспрессии одного аллеля ГКГС класса I или класса II.
15. Способ по любому из пп. 12-14, отличающийся тем, что образцы содержат линии клеток, модифицированные для экспрессии множества аллелей ГКГС класса I или класса II.
16. Способ по любому из пп. 12-15, отличающийся тем, что образцы содержат клеточные линии человека, полученные или извлеченные из множества пациентов.
17. Способ по любому из пп. 12-16, отличающийся тем, что образцы содержат свежие или замороженные образцы опухоли, полученные от множества пациентов.
18. Способ по любому из пп. 12-17, отличающийся тем, что образцы содержат свежие или замороженные образцы ткани, полученные от множества пациентов.
19. Способ по любому из пп. 12-18, отличающийся тем, что образцы содержат пептиды, идентифицированные с использованием T-клеточных анализов.
20. Способ по любому из пп. 12-19, отличающийся тем, что совокупность данных для обучения дополнительно содержит данные, связанные с:
количеством пептидов совокупности пептидов для обучения, присутствующих в образцах;
длиной пептида совокупности пептидов для обучения в образцах.
21. Способ по любому из пп. 12-20, отличающийся тем, что совокупность данных для обучения генерируется путем сравнения совокупности пептидных последовательностей для обучения посредством сравнительного анализа первичной структуры с базой данных, содержащей совокупность известных белковых последовательностей, при этом совокупность последовательностей белков для обучения длиннее, чем и включает пептидные последовательности для обучения.
22. Способ по любому из пп. 12-21, отличающийся тем, что совокупность данных для обучения генерируется на основе выполнения или выполненной масс-спектрометрии на клеточной линии для получения по меньшей мере одного из данных секвенирования пептидов экзома, транскриптома или всего генома из клеточной линии, причем данные секвенирования пептидов содержат по меньшей мере одну белковую последовательность, содержащую изменение.
23. Способ по любому их пп. 12-22, отличающийся тем, что совокупность данных для обучения генерируется на основе получения по меньшей мере одного из стандартных данных нуклеотидного секвенирования экзома, транскриптома и всего генома из образцов нормальной ткани.
24. Способ по любому из пп. 12-23, отличающийся тем, что совокупность данных для обучения дополнительно содержит данные, связанные с последовательностями протеома, связанными с образцами.
25. Способ по любому из пп. 12-24, отличающийся тем, что совокупность данных для обучения дополнительно содержит данные, связанные с последовательностями пептидома ГКГС, связанными с образцами.
26. Способ по любому из пп. 12-25, отличающийся тем, что совокупность данных для обучения дополнительно содержит данные, связанные с измерениями аффинности связывания пептид-ГКГС по меньшей мере для одного из выделенных пептидов.
27. Способ по любому из пп. 12-26, отличающийся тем, что совокупность данных для обучения дополнительно содержит данные, связанные с измерениями стабильности связывания пептид-ГКГС по меньшей мере для одного из выделенных пептидов.
28. Способ по любому из пп. 12-27, отличающийся тем, что совокупность данных для обучения дополнительно содержит данные, связанные с транскриптомами, связанными с образцами.
29. Способ по любому из пп. 12-28, отличающийся тем, что совокупность данных для обучения дополнительно содержит данные, связанные с геномами, связанными с образцами.
30. Способ по любому из пп. 12-29, отличающийся тем, что последовательности пептидов для обучения имеют длину в диапазоне k-меров, где k составляет от 8 до 15 включительно.
31. Способ по любому из пп. 12-30, дополнительно включающий кодирование пептидной последовательности с использованием схемы прямого кодирования (one-hot encoding).
32. Способ по п. 31, дополнительно включающий кодирование пептидных последовательностей для обучения с использованием схемы прямого кодирования слева направо (left-padded one-hot encoding).
33. Способ лечения субъекта, имеющего опухоль, включающий выполнение любого из этапов по пп. 1-32 и дополнительно включающий получение противоопухолевой вакцины, содержащей совокупность выбранных неоантигенов, и введение противоопухолевой вакцины субъекту.
34. Способ изготовления противоопухолевой вакцины, включающий выполнение любого из этапов по пп. 1-33 и дополнительно включающий продуцирование или наличие продуцированной противоопухолевой вакцины, содержащей совокупность выбранных неоантигенов.
35. Противоопухолевая вакцина, содержащая совокупность выбранных неоантигенов по любому из пп. 1-32, выбранных путем выполнения способа по любому из пп. 1-32.
36. Вакцина по п. 35, отличающаяся тем, что противоопухолевая вакцина содержит одно или более из нуклеотидной последовательности, полипептидной последовательности, РНК, ДНК, клетки, плазмиды или вектора.
37. Вакцина по любому из пп. 35, 36, отличающаяся тем, что противоопухолевая вакцина содержит один или более неоантигенов, презентированных на поверхности опухолевой клетки.
38. Вакцина по любому из пп. 35-37, отличающаяся тем, что противоопухолевая вакцина содержит один или более неоантигенов, которые являются иммуногенными у субъекта.
39. Вакцина по любому из пп. 35-38, отличающаяся тем, что противоопухолевая вакцина не содержит один или более неоантигенов, которые индуцируют аутоиммунный ответ против нормальной ткани у субъекта.
40. Вакцина по любому из пп. 35-39, отличающаяся тем, что противоопухолевая вакцина дополнительно содержит адъювант.
41. Вакцина по любому из пп. 35-40, отличающаяся тем, что противоопухолевая вакцина дополнительно содержит наполнитель.
42. Способ по любому из пп. 1-34, отличающийся тем, что выбор совокупности выбранных неоантигенов включает выбор неоантигенов, которые имеют повышенную вероятность быть презентированными на поверхности опухолевой клетки относительно невыбранных неоантигенов на основе модели презентации.
43. Способ по любому из пп. 1-34, 42, отличающийся тем, что выбор совокупности выбранных неоантигенов включает выбор неоантигенов, которые имеют повышенную вероятность быть способными индуцировать опухолеспецифический иммунный ответ у субъекта относительно невыбранных неоантигенов на основе модели презентации.
44. Способ по любому из пп. 1-34, 42, 43, отличающийся тем, что выбор совокупности выбранных неоантигенов включает выбор неоантигенов, которые имеют повышенную вероятность того, что они способны быть презентированы наивным Т-клеткам с помощью профессиональных антигенпрезентирущих клеток (АПК) относительно невыбранных неоантигенов на основе модели презентации, необязательно, при этом АПК представляет собой дендритную клетку (ДК).
45. Способ по любому из пп. 1-34, 42-44, отличающийся тем, что выбор совокупности выбранных неоантигенов включает выбор неоантигенов, которые имеют уменьшенную вероятность быть подвергнутыми ингибированию посредством центральной или периферической толерантности по отношению к невыбранным неоантигенам на основе модели презентации.
46. Способ по любому из пп. 1-34, 42-45, отличающийся тем, что выбор совокупности выбранных неоантигенов включает выбор неоантигенов, которые имеют уменьшенную вероятность быть способными индуцировать аутоиммунный ответ на нормальную ткань у субъекта относительно невыбранных неоантигенов на основе модели презентации.
47. Способ по любому из пп. 1-34, 42-46, отличающийся тем, что данные нуклеотидного секвенирования экзома или транскриптома получают путем проведения секвенирования на опухолевой ткани.
48. Способ по любому из пп. 1-34, 42-47, отличающийся тем, что секвенирование представляет собой секвенирование нового поколения (NGS) или любой подход массивного параллельного секвенирования.
49. Способ по любому из пп. 1-34, 42-48, отличающийся тем, что совокупность числовых вероятностей дополнительно идентифицируют по меньшей мере с ГКГС-аллель-взаимодействующими признаками, включающими по меньшей мере один из следующих:
a. спрогнозированная аффинность, с которой связываются аллель ГКГС и кодируемый неоантигеном пептид;
b. спрогнозированная стабильность комплекса кодируемого неоантигеном пептида-ГКГС;
c. последовательность и длина кодируемого неоантигеном пептида;
d. вероятность презентации кодируемых неоантигеном пептидов с подобной последовательностью в клетках от других индивидуумов, экспрессирующих конкретный аллель ГКГС, как оценивается с помощью масс-спектрометрии протеома или другими способами;
e. уровни экспрессии конкретного аллеля ГКГС у исследуемого субъекта (например, измеренного с помощью РНК-секвенирования или масс-спектрометрии);
f. в целом независимая от последовательности пептида кодируемого неоантигеном вероятность презентации конкретным аллелем ГКГС у других отличных субъектов, которые экспрессируют конкретный аллель ГКГС;
g. в целом независимая от последовательности пептида кодируемого неоантигеном вероятность презентации аллелями ГКГС в одном и том же семействе молекул (например, HLA-A, HLA-B, HLA-C, HLA-DQ, HLA-DR, HLA-DP) у других отличных субъектов.
50. Способ по любому из пп. 1-34, 42-49, отличающийся тем, что совокупность числовых значений вероятностей дополнительно идентифицируют по меньшей мере по ГКГС-аллель-невзаимодействующим признакам, включающим по меньшей мере один из следующих:
a. С- и N-концевые последовательности, фланкирующие кодируемый неоантигеном пептид в его исходной белковой последовательности;
b. присутствие мотивов расщепления протеазой в кодируемом неоантигеном пептиде, необязательно оцененном в соответствии с экспрессией соответствующих протеаз в опухолевых клетках (как измерено с помощью РНК-секвенирования или масс-спектрометрии);
c. скорость обмена исходного белка, измеренная в соответствующем типе клеток;
d. длина исходного белка, необязательно, учитывая конкретные варианты сплайсинга («изоформы»), наиболее высоко экспрессируемые в опухолевых клетках, как измерено с помощью РНК-секвенирования или масс-спектрометрии протеома, или как спрогнозировано из аннотации мутаций зародышевой линии или соматических мутаций сплайсинга, обнаруженных в данных последовательностей ДНК или РНК;
e. уровень экспрессии протеасомы, иммунопротеасомы, тимопротеасомы или других протеаз в опухолевых клетках (которые могут быть измерены с помощью РНК-секвенирования, масс-спектрометрии протеома или иммуногистохимии);
f. экспрессия исходного гена кодируемого неоантигеном пептида (например, как измерено с помощью РНК-секвенирования или масс-спектрометрии);
g. типичная тканеспецифическая экспрессия исходного гена кодируемого неоантигеном пептида на разных стадиях клеточного цикла;
h. полный каталог признаков исходного белка и/или его доменов, как можно найти, например, в uniProt или PDB http://www.rcsb.org/pdb/home/home.do;
i. признаки, описывающие свойства домена исходного белка, содержащего пептид, например: вторичную или третичную структуру (например, альфа-спираль против бета-складчатости); альтернативный сплайсинг;
j. вероятность презентации пептидов из исходного белка исследуемого кодируемого неоантигеном пептида у других отличных субъектов;
k. вероятность того, что пептид не будет обнаружен или сверхпрезентирован при масс-спектрометрии из-за технических погрешностей;
l. экспрессия различных генных модулей/путей, измеренных при помощи РНК-секвенирования (которые не обязательно содержат исходный белок пептида), которые являются информативными относительно состояния опухолевых клеток, стромы или инфильтрирующих опухоли лимфоцитов (TIL);
m. число копий исходного гена кодируемого неоантигеном пептида в опухолевых клетках;
n. вероятность того, что пептид связывается с ТАР, или измеренную или спрогнозированную аффинность связывания пептида с ТАР;
o. уровень экспрессии ТАР в опухолевых клетках (который может быть измерен с помощью РНК-секвенирования, масс-спектрометрии протеома, иммуногистохимии);
p. присутствие или отсутствие опухолевых мутаций, включая, но не ограничиваясь ими:
i. драйверные мутации в известных генах-драйверах рака, такие как EGFR, KRAS, ALK, RET, ROS1, ТР53, CDKN2A, CDKN2B, NTRK1, NTRK2, NTRK3;
ii. в генах, кодирующих белки, участвующие в механизмах презентации антигенов (например, В2М, HLA-A, HLA-B, HLA-C, ТАР-1, ТАР-2, ТАРВР, CALR, CNX, ERP57, HLA-DM, HLA-DMA, HLA-DMB, HLA-DO, HLA-DOA, HLA-DOBHLA-DP, HLA-DPA1, HLA-DPB1, HLA-DQ, HLA-DQA1, HLA-DQA2, HLA-DQB1, HLA-DQB2, HLA-DR, HLA-DRA, HLA-DRB1, HLA-DRB3, HLA-DRB4, HLA-DRB5 или любой из генов, кодирующих компоненты протеасомы или иммунопротеасомы), пептиды, чья презентация опирается на компонент механизма презентации антигена, который подвержен мутации с утратой функции в опухоли, имеют более низкую вероятность презентации;
q. присутствие или отсутствие функциональных полиморфизмов зародышевой линии, включая, но не ограничиваясь:
i. в генах, кодирующих белки, участвующие в механизмах презентации антигенов (например, В2М, HLA-A, HLA-B, HLA-C, ТАР-1, ТАР-2, ТАРВР, CALR, CNX, ERP57, HLA-DM, HLA-DMA, HLA-DMB, HLA-DO, HLA-DOA, HLA-DOBHLA-DP, HLA-DPA1, HLA-DPB1, HLA-DQ, HLA-DQA1, HLA-DQA2, HLA-DQB1, HLA-DQB2, HLA-DR, HLA-DRA, HLA-DRB1, HLA-DRB3, HLA-DRB4, HLA-DRB5 или в любом из генов, кодирующих компоненты протеасомы или иммунопротеасомы);
r. тип опухоли (например, НМРЛ, меланома);
s. клинический подтип опухоли (например, плоскоклеточный рак легкого в противовес неплоскоклеточного);
t. анамнез курения;
u. типичная экспрессия исходного гена пептида в соответствующем типе или клиническом подтипе опухоли, необязательно стратифицированном по драйверной мутации.
51. Способ по любому из пп. 1-34, 42-50, отличающийся тем, что по меньшей мере одна мутация представляет собой инсерционно-делеционную мутацию со сдвигом или без сдвига рамки считывания, миссенс- или нонсенс-замену, изменение сайта сплайсинга, геномную перестройку или слияние генов или любые изменения генома или экспрессии, приводящие к нео-ОРС.
52. Способ по любому из пп. 1-34, 42-51, отличающийся тем, что опухолевая клетка выбирается из группы, состоящей из: рака легкого, меланомы, рака молочной железы, рака яичников, рака предстательной железы, рака почки, рака желудка, рака толстой кишки, рака яичек, рака головы и шеи, рака поджелудочной железы, рака мозга, B-клеточной лимфомы, острого миелогенного лейкоза, хронического миелогенного лейкоза, хронического лимфолейкоза и T-клеточного лимфоцитарного лейкоза, немелкоклеточного рака легкого и мелкоклеточного рака легкого.
53. Способ по любому из пп. 1-34, 42-52, дополнительно включающий получение противоопухолевой вакцины, содержащей совокупность выбранных неоантигенов или ее подсовокупность, необязательно дополнительно включающий введение противоопухолевой вакцины субъекту.
54. Способ по любому из пп. 1-34, 42-53, отличающийся тем, что по меньшей мере один из неоантигенов в совокупности выбранных неоантигенов, когда он находится в полипептидной форме, содержит по меньшей мере одно из: аффинности связывания с ГКГС с величиной ИК50 менее 1000 нМ для полипептидов ГКГС 1-ого класса длиной 8-15, 8, 9, 10, 11, 12, 13, 14 или 15 аминокислот, присутствие мотивов последовательности внутри или рядом с полипептидом в исходной белковой последовательности, способствующей расщеплению протеасомой, и присутствие мотивов последовательности, способствующих ТАР транспорту.
55. Способ для генерации модели для идентификации одного или более неоантигенов, которые имеют вероятность быть презентированными на поверхности опухолевой клетки опухолевой клетки, включающий выполнение этапов:
получения данных масс-спектрометрии, содержащих данные, связанные с множеством выделенных пептидов, элюированных из главного комплекса гистосовместимости (ГКГС), полученного из множества образцов;
получения совокупности данных для обучения путем по меньшей мере идентификации совокупности пептидных последовательностей для обучения, присутствующих в образцах, и одного или более ГКГС, связанных с каждой пептидной последовательностью для обучения;
обучение совокупности числовых параметров модели презентации с использованием совокупности данных для обучения, содержащей пептидные последовательности для обучения, модели презентации, обеспечивающей множество числовых значений вероятностей, что пептидные последовательности из опухолевой клетки презентированы одним или более аллелями ГКГС на поверхности опухолевых клеток.
56. Способ по п. 55, отличающийся тем, что модель презентации представляет зависимость между:
присутствием определенной аминокислоты в определенном положении пептидной последовательности; и
вероятностью презентации одним из аллелей ГКГС опухолевой клетки пептидной последовательности, содержащей определенную аминокислоту в конкретном положении.
57. Способ по любому из пп. 55, 56, отличающийся тем, что образцы содержат линии клеток, модифицированные для экспрессии одного аллеля ГКГС класса I или класса II.
58. Способ по любому из пп. 55-57, отличающийся тем, что образцы содержат линии клеток, модифицированные для экспрессии множества аллелей ГКГС класса I или класса II.
59. Способ по любому из пп. 55-58, отличающийся тем, что образцы содержат клеточные линии человека, полученные или извлеченные из множества пациентов.
60. Способ по любому из пп. 55-59, отличающийся тем, что образцы содержат свежие или замороженные образцы опухоли, полученные от множества пациентов.
61. Способ по любому из пп. 55-60, отличающийся тем, что образцы содержат пептиды, идентифицированные с использованием T-клеточных анализов.
62. Способ по любому из пп. 55-61, отличающийся тем, что совокупность данных для обучения дополнительно содержит данные, связанные с:
количеством пептидов совокупности пептидов для обучения, присутствующих в образцах;
длиной пептида совокупности пептидов для обучения в образцах.
63. Способ по любому из пп. 55-62, отличающийся тем, что получение совокупности данных для обучения включает:
получение совокупности белковых последовательностей для обучения на основе пептидных последовательностей для обучения путем сравнения совокупности пептидных последовательностей для обучения посредством сравнительного анализа первичной структуры с базой данных, содержащей совокупность известных белковых последовательностей, при этом совокупность белковых последовательностей для обучения длиннее и включают пептидные последовательности для обучения.
64. Способ по любому из пп. 55-63, отличающийся тем, что получение совокупности данных для обучения включает:
выполнение или выполненную масс-спектрометрию на клеточной линии для получения по меньшей мере одного из данных нуклеотидного секвенирования экзома, транскриптома или всего генома из клеточной линии, данные нуклеотидного секвенирования, содержащие по меньшей мере одну последовательность белка, содержащую мутацию.
65. Способ по любому из пп. 55-64, отличающийся тем, что обучение совокупности параметров модели презентации включает:
кодирование пептидных последовательностей для обучения с использованием схемы прямого кодирования (one-hot encoding).
66. Способ по любому из пп. 55-65, дополнительно включающий:
получение по меньшей мере одного из стандартных данных нуклеотидного секвенирования экзома, транскриптома и всего генома из образцов нормальной ткани; и
обучение совокупности параметров модели презентации с использованием данных стандартного нуклеотидного секвенирования.
67. Способ по любому из пп. 55-66, отличающийся тем, что совокупность данных для обучения дополнительно содержит данные, связанные с последовательностями протеома, связанными с образцами.
68. Способ по любому из пп. 55-67, отличающийся тем, что совокупность данных для обучения дополнительно содержит данные, связанные с последовательностями пептидома ГКГС, связанными с образцами.
69. Способ по любому из пп. 55-68, отличающийся тем, что совокупность данных для обучения дополнительно содержит данные, связанные с измерениями аффинности связывания пептид-ГКГС для по меньшей мере одного из выделенных пептидов.
70. Способ по любому из пп. 55-69, отличающийся тем, что совокупность данных для обучения дополнительно содержит данные, связанные с измерениями стабильности связывания пептид-ГКГС для по меньшей мере одного из выделенных пептидов.
71. Способ по любому из пп. 55-70, отличающийся тем, что совокупность данных для обучения дополнительно содержит данные, связанные с транскриптомами, связанными с образцами.
72. Способ по любому из пп. 55-71, отличающийся тем, что совокупность данных для обучения дополнительно содержит данные, связанные с геномами, связанными с образцами.
73. Способ по любому из пп. 55-72, отличающийся тем, что обучение совокупности числовых параметров дополнительно включает:
логическое регрессирование совокупности параметров.
74. Способ по любому из пп. 55-73, отличающийся тем, что последовательности пептидов для обучения имеют длину в диапазоне k-меров, где k составляет от 8 до 15 включительно.
75. Способ по любому из пп. 55-74, отличающийся тем, что обучение совокупности числовых параметров модели презентации включает:
кодирование пептидных последовательностей для обучения с использованием схемы прямого кодирования слева направо (left-padded one-hot encoding).
76. Способ по любому из пп. 55-75, отличающийся тем, что обучение совокупности числовых параметров дополнительно включает:
определение значений для совокупности параметров с использованием алгоритма глубокого обучения (deep learning).
77. Способ для генерации модели для идентификации одного или более неоантигенов, которые имеют вероятность быть презентированными на поверхности опухолевой клетки опухолевой клетки, включающий выполнение этапов:
получения данных масс-спектрометрии, содержащих данные, связанные с множеством выделенных пептидов, элюированных из главного комплекса гистосовместимости (ГКГС), полученного из множества свежих или замороженных образцов опухоли;
получения совокупности данных для обучения путем по меньшей мере идентификации совокупности пептидных последовательностей для обучения, присутствующих в образцах опухоли и презентированных на одном или более аллелях ГКГС, связанных с каждой пептидной последовательностью для обучения;
получения совокупности белковых последовательностей для обучения на основе пептидных последовательностей для обучения; и
обучение совокупности числовых параметров модели презентации с использованием белковых последовательностей для обучения и пептидных последовательностей для обучения, модели презентации, обеспечивающей множество числовых значений вероятностей, что пептидные последовательности из опухолевой клетки презентированы одним или более аллелями ГКГС на поверхности опухолевых клеток.
78. Способ по п. 77, отличающийся тем, что модель презентации представляет зависимость между:
присутствием пары определенного одного из аллелей ГКГС и определенной аминокислоты в определенном положении пептидной последовательности; и
вероятностью презентации на поверхности опухолевой клетки, одним определенным ГКГС аллелем указанной пары, такой пептидной последовательности, содержащей определенную аминокислоту в определенном положении.
| POLYAKOVA A | |||
| et al., Proteogenomics meets cancer immunology: mass spectrometric discovery and analysis of neoantigens, Expert review of proteomics, 2015, V | |||
| Способ гальванического снятия позолоты с серебряных изделий без заметного изменения их формы | 1923 |
|
SU12A1 |
| Кипятильник для воды | 1921 |
|
SU5A1 |
| WO 2011143656, 17.11.2011 | |||
| FRITSCH E | |||
| F | |||
| et al., HLA-binding properties of tumor neoepitopes in humans, Cancer immunology research, 2014, V | |||
| Аппарат для очищения воды при помощи химических реактивов | 1917 |
|
SU2A1 |
| Приспособление для точного наложения листов бумаги при снятии оттисков | 1922 |
|
SU6A1 |
Авторы
Даты
2020-08-04—Публикация
2016-12-16—Подача