Изобретение относится к биотехнологии, медицине, молекулярной биологии и вирусологии и позволяет произвести пробоподготовку библиотек, содержащих целевые фрагменты генов вируса гепатита В (ВГВ) методом высокопроизводительного секвенирования, для целей поиска мутаций вируса гепатита В как при решении задач эпидемиологического надзора, так и в научно-исследовательских целях.
ВГВ представляет собой ДНК-содержащий вирус из семейства гепаднавирусов и является возбудителем вирусного гепатита В. В мире по различным оценкам от 3% до 6% людей инфицировано вирусом гепатита В. По данным экспертных оценок, в РФ насчитывается около 3 млн пациентов с хроническим гепатитом В. Согласно Аналитическому обзору Вирусных гепатитов в Российской Федерации под редакцией академика РАН, профессора В.И. Покровского и академика РАН, профессора А.А. Тотоляна, ежегодная смертность от ВГВ в России составляет примерно 2 тысячи человек [Вирусные гепатиты в Российской Федерации. Аналитический обзор. 11 выпуск / Под ред. В.И. Покровского, А.А. Тотоляна. - СПб.: ФБУН НИИЭМ имени Пастера, 2018. - 112 с].
Данные многочисленных исследований говорят о том, что генотип вируса гепатита В может оказывать влияние на течение заболевания. В настоящее время ВГВ подразделяют на 10 генотипов (А-J), отличающихся по составу нуклеотидных последовательностей более чем на 8%. Изучение генетической гетерогенности ВГВ стало основным направлением в исследованиях, направленных на понимание взаимосвязи между геновариантами вируса и широким спектром клинических и патологических состояний, связанных с инфекцией, ответом на антивирусную терапию и прогнозом заболевания [Araujo NM, Waizbort R, Kay A. Hepatitis B virus infection from an evolutionary point of view: how viral, host, and environmental factors shape genotypes and subgenotypes. Infect Genet Evol. 2011 Aug; 11(6):1199-207].
Например, было показано, что подтип А1, характерный для Южной и Восточной Африки, отличается быстрой сероконверсией и низкой вирусной нагрузкой, но при этом наблюдается высокая частота возникновения гепатоцеллюлярной карциномы [Kramvis, A., Kew, M.C., 2007. Molecular characterization of subgenotype A1 (subgroup Aa) of hepatitis B virus. Hepatol. Res. 37, S27-S32]. Генотип С, который превалирует в Юго-Восточной Азии, отличается повышенным риском быстрого развития цирроза печени и гепатоцеллюлярной карциномы [Chan, H.L., Hui, A.Y., Wong, M.L., Tse, A.M., Hung, L.C., Wong, V.W., Sung, J.J., 2004. Genotype C hepatitis B virus infection is associated with an increased risk of hepatocellular carcinoma. Gut 53, 1494-1498; Chen, C.H., Eng, H.L., Lee, Kuo, F.Y., Lu, S.N., Huang, C.M., Tung, H.D., Chen, C.L., Changchien, C.S., 2004. Correlations between hepatitis B virus genotype and cirrhotic or non-cirrhotic hepatoma. Hepatogastroenterology 51, 552-555]. В России наиболее распространен генотип D, при котором отмечается более низкая частота встречаемости ГЦК, чем для других подтипов [Araujo NM, Waizbort R, Kay A. Hepatitis B virus infection from an evolutionary point of view: how viral, host, and environmental factors shape genotypes. Infect Genet Evol. 2011 Aug; 11(6):1199-207; Останкова Ю.В., Семенов А.В., Зуева Е.Б., Тотолян Арег А. Первые случаи выявления вируса гепатита В субгенотипа D4 у больных хроническим, острым и скрытым вирусным гепатитом В в Российской Федерации. Молекулярная генетика, микробиология и вирусология. 2020;38(4):180-187].
Препараты для лечения вирусного гепатита В относятся к двум группам: противовирусные препараты прямого действия (ПППД) и иммуномодуляторы, например интерферон альфа-2а [Кузнецов Н.И. (2012). Вирусный гепатит в. Российский семейный врач, 16(4), 13-18]. ПППД представляют собой аналоги нуклеотидов/нуклеозидов, которые ингибируют обратную транскриптазу ВГВ, и рекомендованы ВОЗ в качестве терапии первой линии [Guidelines for the prevention, care and treatment of persons with chronic hepatitis B infection. World Health Organization. ISBN 9789241549059 (NLM classification: WC 536)].
Известен ряд мутаций вируса гепатита В в RT-домене Р-гена, которые приводят к резистентности вируса к терапии аналогами нуклеотидов/нуклеозидов. В частности, мутации L80I\V, V173L, L180M, A181T, T184S, S202G и M204I\V ассоциированы с резистентностью к терапии ламивудином (нуклеозидный ингибитор обратной транскриптазы), а мутации A181S/Т/V и N236T ассоциированы с резистентностью к терапии адефовиром (нуклеотидный ингибитор обратной транскриптазы) [Rhee SY, Margeridon-Thermet S, Nguyen MH, Liu TF, Kagan RM, Beggel B, Verheyen J, Kaiser R, Shafer RW. Hepatitis B virus reverse transcriptase sequence variant database for sequence analysis and mutation discovery. Antiviral Res. 2010 Dec; 88(3):269-75; Nakajima S, Watashi K, Kato T, Muramatsu M, Wakita T, Tamura N, Hattori SI, Maeda K, Mitsuya H, Yasutake Y, Toyoda T. Biochemical and Structural Properties of Entecavir-Resistant Hepatitis B Virus Polymerase with L180M/M204V Mutations. J Virol. 2021 Jil 26;95(16):е0240120].
Данные мутации настолько важны, что существуют коммерческие наборы для их обнаружения методом ПЦР и последующего секвенирования по Сэнгеру, например, набор АмплиСенс® HBV-Resist-Seg (https://goszdravnadzor.ru/ru-roszdravnadzora-%E2%84%96-rzn-2016-4228-ot-17-oktyabrya-2022-goda/). Помимо RT-домена Р-гена остальные регионы генома ВГВ также вызывают интерес. В нескольких работах было показано, что Х-ген ВГВ участвует в канцерогенезе и что мутации в данном регионе могут быть связаны с клиническими особенностями возникновения и развития гепатоцеллюлярной карциномы [Malik A, Singhal DK, Albanyan A, Husain SA, Kar P. Hepatitis B virus gene mutations in liver diseases: a report from New Delhi. PLoS One. 2012;7(6):е39028; Al-Qahtani AA, Al-Anazi MR, Nazir N, Ghai R, Abdo AA, Sanai FM, Al-Hamoudi WK, Alswat KA, AL-Ashgar HI, Khan MQ, Albenmousa A, Cruz DD, Bohol MFF, Al-Ahdal MN. Hepatitis B virus (HBV) X gene mutations and their association with liver disease progression in HBV-infected patients. Oncotarget. 2017 Nov 6;8(62):105115-105125]. Таким образом существует явная потребность в определении полногеномной последовательности ВГВ.
Ранее в большинстве исследований подтип ВГВ определялся при помощи ПЦР и секвенированием по Сэнгеру небольших участков генома, либо серологически. В исследованиях полной нуклеотидной последовательности ВГВ, как правило, используется метод вложенной ПЦР (nested PCR) и последующего секвенирования амплифицированных участков по Сэнгеру.
Например, в исследовании, проведенном в 2011 году в Иране, таким способом было секвенировано 50 полных геномов ВГВ [Garmiri P, Rezvan H, Abolghasemi H, Allain JP. Full genome characterization of hepatitis B virus strains from blood donors in Iran. J Med Virol. 2011 1ип;83(6):948-52]. Данный метод был использован и в России в 2020 году для секвенирования 3 полных генов ВГВ [Останкова Ю.В., Семенов А.В., Зуева Е.Б., Тотолян Арег А. Первые случаи выявления вируса гепатита В субгенотипа D4 у больных хроническим, острым и скрытым вирусным гепатитом В в Российской Федерации. Молекулярная генетика, микробиология и вирусология. 2020;38(4):180-187].
В 2011 году в Чили для секвенирования 21 образца ВГВ был использован схожий подход - амплификация полного генома и последующее секвенирование участков ампликонов с помощью различных праймеров [Venegas M, Alvarado-Mora MV, Villanueva RA< Rebello Pinho JR, Carrilho FJ, Locarnini S, Yuen L, Brahm J. Phylogenetic analysis of hepatitis B virus genotype F complete genome sequences from Chilean patients with chronic infection. J Med Virol. 2011 Sep;83(9):1530-6].
В 2022 году в Тунисе было проведено исследование, в котором были определены полноразмерные геномы ВГВ у 5 пациентов с хроническим гепатитом до и во время долгосрочной терапии, использовалось секвенирование по Сэнгеру [Belaiba Z, Ayouni K, Gdoura M, Kammoun Rebai W, Touzi H, Sadraoui A, Hammemi W, Yacoubi L, Abdelati S, Hamzaoui L, Msaddak Azzouz M, Chouikha A, Triki H. Whole genome analysis of hepatitis B virus before and during long-term therapy in chronic infected patients: Molecular characterization, impact on treatment and liver disease progression. Front Microbiol. 2022 Oct 17; 13:1020147].
Высокопроизводительное секвенирование (NGS) в настоящее время является наиболее оптимальным методом полногемного секвенирования вируса гепатита В с точки зрения соотношения получаемой информации, стоимости и времени анализа. В 2020 году было опубликовано исследование, выполненное в Бразилии, в котором описывалось секвенирование 5 полных геномов ВГВ при помощи NGS, при этом авторы использовали набор Nextera Illumina для тагментации ампликонов [Hebeler-Barbosa F, Wolf IR, Valente GT, Mello FCDA, Lampe E, Pardini MIMC, Grotto RMT. A New Method for Next-Generation Sequencing of the Full Hepatitis B Virus Genome from A Clinical Specimen: Impact for Virus Genotyping. Microorganims. 2020 Sep 11;8(9):1391].
В 2023 году в США были определены 20 полных геномов ВГВ, при этом в качестве матрицы использовалась РНК ВГВ, пробоподготовка осуществлялась при помощи наборов Ovation® RNA-Seq System V2 NuGEN и TruSeq RNAseq Illumina [Chang S, Hedskog C, Parhy B, Martin R, Mo H, Maiorova E, Zoulim F. Sequence characterization of extracellular HBV RNA in patient plasma. J Viral Hepat. 2023 Jan;30(1):29-38].
Из уровня техники известен зонд для обнаружения вируса гепатита В и способ обнаружения места внедрения вируса гепатита В с высокой эффективностью, основанный на методе анализа секвенирования следующего поколения с использованием зонда [заявка US 20210130916, дата публикации 05.06.2021], где применяя зонд к методу анализа секвенирования следующего поколения, можно проанализировать сайты внедрения HBV в геноме человека с низкими затратами и высокой эффективностью. Данное решение не позволяет определять мутации вируса и ограничен одной популяцией человека.
В настоящее время в мире существует крайне ограниченное количество панелей NGS для секвенирования ВГВ. В качестве примеров можно привести:
- DeepChek®Assay-HBV RT Genotyping and Drug Resistance V1 (RUO) компании Advanced Biological Laboratories (ABL) S.A. (Люксембург) [https://wwww.ablsa.com/laboratory-applications/deepchek-hbv-rt-genotyping-dr-assay]. Данный набор позволяет секвенировать не весь геном ВГВ, а только RT-домен P-гена. Секвенирование наработанных ампликонов возможно как на платформе Illumina, так и по методу Сэнгера;
- Набор CDCAP® HBV Capture Kit от компании CD Genomics (США) [https://www.cd-genomics.com/diseasepanel/products_71.html]. Данный набор представляет собой гибридизационную панель и предполагает секвенирование на платформе Illumina. Из-за гибридизационного обогащения данный набор требует для анализа не менее чем 500 нг ДНК ВГВ, что является значительным количеством и вызывает опредленные сложности в их получении. Кроме того, для данного набора требуются образцы с высокой вирусной нагрузкой.
Таким образом, существует необходимость в получении эффективных и простых способов приготовления библиотек для целевого секвенирования вируса гепатита В с возможностью кастомизации, т.е. изменения праймеров в панели для задач конкретного исследования.
Технической задачей, на решение которой направлено заявляемое решение, является разработка эффективного способа пробоподготовки для NGS-секвенирования полного генома ВГВ, содержащего известные эпидемиологически и клинически значимые мутации.
Заявляемое изобретение позволяет преодолеть недостатки предшествующего уровня техники и в значительной мере уменьшает трудовые и финансовые затраты при пробоподготовке образцов для секвенирования NGS.
Технический результат достигается за счет таргетного (целевого) секвенирования участков генома вируса гепатита В, в сумме составляющих весь его геном. Полногеномное секвенирование ВГВ позволяет определить подтип вируса и мутации, представляющие клинический или научный интерес. Посредством разработанных олигонуклеотидных праймеров, имеющих адаптерные подпоследовательности в своем составе, возможна амплификация целевых участков генома, при этом не требуется проводить дорогостоящий этап лигирования адаптеров, используемый в ряде протоколов. При этом амплификацию проводят с использованием двух пулов олигонуклеотидных праймеров, где для первого пула применяют олигонуклеотидные праймеры, имеющие структуру SEQ ID NO: 1-30, для второго пула применяют олигонуклеотидные праймеры, имеющие структуру SEQ ID NO: 31 - 55. Амплифицированные фрагменты ДНК ВГВ, после проведения секвенирования, позволяют определить полногеномную последовательность вируса гепатита В и обнаружить значимые мутации в исследуемом образце.
Сложность выбора праймеров обусловлена требованием к их строгой видоспецифичности, необходимости обеспечить амплификацию участков ДНК ВГВ для всех известных подтипов данного вируса и отсутствие наработки ампликонов по матрице ДНК человека. Амплификация в формате мультиплекса накладывает дополнительные ограничения на праймеры, требуется обеспечить отсутствие кросс-комплементарности праймеров между собой и используемыми адаптерами.
Предложенные в изобретении синтетические олигонуклеотидные праймеры для полногеномного секвенирования ВГВ методом NGS имеют следующую структуру: SEQ ID NO: 1-55. При этом олигонуклеотидные праймеры, имеющие структуры SEQ ID NO: 1, 3, 5, 8, 10, 12, 15, 16, 18, 22-24, 27, 28, 31, 34, 36, 38, 40, 42, 45, 47, 48, 51, 54 выполняют функции прямых праймеров, а олигонуклеотидные праймеры, имеющие структуры SEQ ID NO: 2, 4, 6, 7, 9, 11, 13, 14, 17, 19-21, 25, 26, 29, 30, 32, 33, 35, 37, 39, 41, 43-44, 46, 49, 50, 52, 53, 55 выполняют функции обратных праймеров.
Кроме того, олигонуклеотидные праймеры содержат в своем составе универсальные адаптерные последовательности, представленные в перечне последовательностей как SEQ ID NO: 56 и SEQ ID NO: 57, которые в дальнейшем значительно упрощают индексацию продуктов амплификации. Адаптерные последовательности SEQ ID NO: 56 расположены на концах прямых праймеров, а SEQ ID NO: 57 расположены на концах обратных праймеров.
При разработке праймеров, применяемых для таргетной амплификации фрагментов вируса гепатита В в заявляемом способе, олигонуклеотидные последовательности были подобраны в полуавтоматическом режиме с учетом имеющейся информации о распространенных подтипах вируса гепатита В. Данный процесс включал в себя загрузку последовательностей геномов ВГВ из баз данных NCBI [https://www.ncbi.nlm.nih.gov/] и HBVdb [https://hbvdb.lyon.inserm.fr], их выравнивание с целью определения консервативных фрагментов и, непосредственно, генерацию праймерных последовательностей. Температуры плавления полученных олигонуклеотидов определялись с помощью инструмента Multiple Primer Analyzer от Thermo Fisher Scientific (США). Подбор каждой новой праймерной пары в пуле осуществлялся таким образом, чтобы максимизировать сумму изменений термодинамических потенциалов Гиббса попарных взаимодействий всех последовательностей этого пула на данном шаге. Расчет термодинамических потенциалов проводился с помощью утилиты PrimerDimer. При этом учитывались дополнительные адаптерные части, добавляемые к праймерам на этапе синтеза, значительно удешевляющие и ускоряющие процесс индексации продуктов амплификации. Затем, с помощью программы blastn оценивалась специфичность каждой полученной последовательности ко всем известным организмам, в частности, Homo sapiens. Описанные действия позволяют минимизировать неспецифическое взаимодействие между праймерами и участками ДНК человека или других организмов. Длины ампликонов были выбраны в пределах 300 п.н., что позволяет избежать этапа отбора фрагментов с заданной длиной и добиться совместимости с большинством наборов реагентов для секвенирования от компании Illumina.
Полученные олигонуклеотиды на одном конце имеют праймерную последовательность, комплементарную участкам генома вируса гепатита В, а на другом - адаптерные последовательности, следовательно, полученные после амплификации целевые фрагменты сразу фланкированы такими адаптерными последовательностями.
Для индексации используются олигонуклеотиды, несущие различные индексы, которые позволяют отличать данные секвенирования молекул из разных образцов. Примеры подобных индексных последовательностей доступны на сайтах производителей наборов реагентов для высокопроизводительного секвенирования, например индексы Nextera [https://support-docs.illumina.com/SHARE/AdapterSeq/Content/SHARE/AdapterSeq/Nextera/DNAIndexesNXT.htm]. Олигонуклеотиды, используемые для индексации, в свою очередь, комплементарны адаптерным последовательностям с одного конца и соответствуют индексам с другого.
Так, для реализации заявляемого решения использовались известные олигонуклеотиды со следующей структурой:
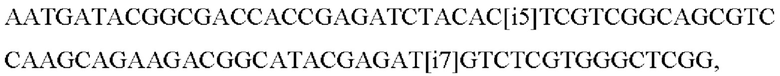
которые используются для подготовки библиотек для секвенирования на платформе Illumina [https://dnatech.genomecenter.ucdavis.edu/wp-content/uploads/2019/03/illumina-adapter-sequences-2019-1000000002694-10.pdf].
Таким образом, в процессе индексации происходят этапы отжига олигонуклеотидов на концах целевых фрагментов и амплификации. После этого продукты амплификации подвергаются секвенированию методом NGS на платформе Illumina. В настоящее время такие подходы широко распространены при клиническом секвенировании полного экзома человека, но реже применяются к патогенным микроорганизмам.
По сравнению с известным протоколом пробоподготовки NEBNext® UlrTM II DNA Library Prep (New England Biolabs) [https://intarnational.neb.com/products/e7645-nebnext-ultra-ii-dna-library-prep-kit-for-illumina], предлагаемый способ пробоподготовки позволяет избежать дорогостоящих этапов ферментативного или ультразвукового фрагментирования ампликонов, фосфорилирования концов ампликонов и лигирования адаптеров, наработанные ампликоны имеют подходящую для секвенирования длину и содержат адаптерные последовательности для дальнейшей индексации.
По сравнению с известным протоколом пробоподготовки NextraXT DNA Library Preparation Kit (New England Biolabs) [https://www.illumina.com/products/by-type/sequencing-kits/library-prep-kits/nextera-xt-dna.html], предлагаемый способ пробоподготовки позволяет избежать дорогостоящего этапа тагментации, наработанные ампликоны имеют подходящую для секвенирования длину и содержат адаптерные последовательности для дальнейшей индексации, что существенно упрощает и удешевляет анализ полученных при секвенировании данных.
Заявляемое изобретение является результатом работы в рамках исследований, выполненных в ФБУН ЦНИИ Эпидемиологии Роспотребнадзора (Москва, Россия). Представленный способ был отработан более чем на 400 образцах биологического материала, представляющих собой ДНК, выделенную из плазмы крови больных вирусным гепатитом В при помощи набора «РИБО-преп» (AmpliSens, Россия) согласно рекомендациям производителя. Полученная панель была опробована на образцах с вирусной нагрузкой от 103 до 107 МЕ/мл.
Для выделения ДНК может быть использован комплект реагентов «РИБО-преп» (АмплиСенс, Россия) или любой аналогичный коммерческий набор для выделения ДНК. Материалом для исследования является ДНК, выделенная из образцов плазмы или сыворотки крови. Для анализа одного образца требуется 20 мкл ДНК, выделенной из 100 мкл образца плазмы или сыворотки крови с вирусной нагрузкой не менее 103 МЕ/мл. Две реакции амплификации проводят с использованием ДНК в качестве матрицы и двух пулов специфических олигонуклеотидов. Первый пул олигонуклеотидов содержит олигонуклеотиды SEQ ID NO: 1-30, второй пул олигонуклеотидов содержит олигонуклеотиды SEQ ID NO: 31-55 с концентрацией в реакционной смеси, указанной в Таблице 1. При этом на концах прямых праймеров расположены адаптерные последовательности SEQ ID NO: 56, а концах обратных праймеров расположены адаптерные последовательности SEQ ID NO: 57.
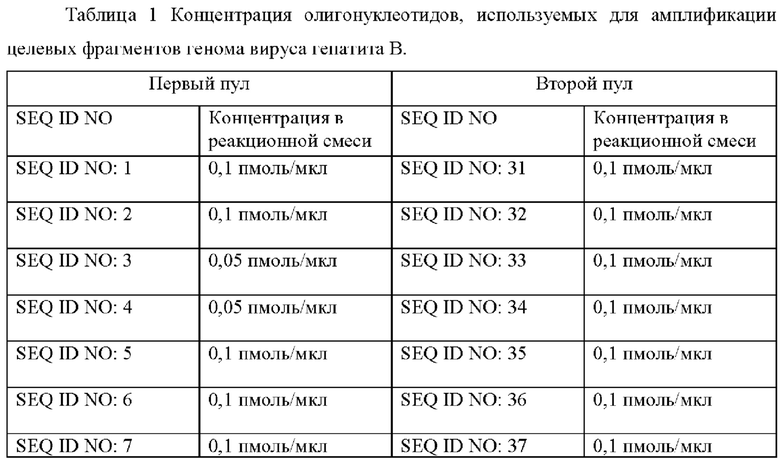
ПЦР-микс объемом 15 мкл на одну реакцию содержит 10 мкл ПЦР-смеси-2 blue (AmpliSens, Россия), 1,4 мкл эквимолярной смеси четырех дезоксирибонуклеозидтрифосфатов со стоковой концентрацией 4,4 мМ, воду milli-Q и заявляемые олигонуклеотиды в указанных в Таблице 1 концентрациях.
ДНК-матрицу добавляют в количестве 10 мкл, итоговый объем реакции составляет 25 мкл. Продуктами амплификации являются целевые участки ДНК вируса гепатита В. Длины получаемых фрагментов составляют 128-296 п.н. без учета длин адаптерных и индексных последовательностей, что обеспечивает их полное покрытие при использовании наборов реагентов для секвенирования MiSeq Reagent Kit v2 (300-cycles), MiSeq Reagent Kit v3 (600-cycle), HiSeq Rapid SBS Kit v2 (500 cycles) Illumina (США). Температурный профиль амплификации для получения ПНР-продукта указан в Таблице 2.
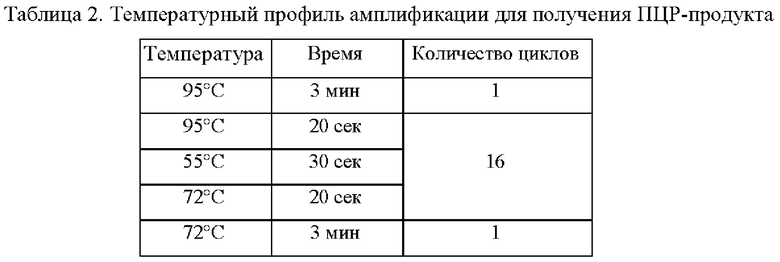
Синтезированные олигонуклеотиды на одном конце уже содержат праймерную последовательность, комплементарную целевым участкам генов, а на другом - адаптерные последовательности двух типов: для прямых праймеров - SEQ ID NO: 56, для обратных праймеров - SEQ ID NO: 57. Таким образом, полученные после амплификации целевые фрагменты сразу фланкируются адаптерными последовательностями, что существенно ускоряет и удешевляет пробоподготовку.
Затем проводят очистку ПЦР-продуктов от реакционной смеси с использованием магнитных частиц, например, AMPure XP beads (Beckman Coulter, США) в объемном отношении 1:1.
После этого осуществляют индексацию полученных фрагментов. ПЦР-микс объемом 20 мкл на одну реакцию содержит 10 мкл ПЦР-смеси-2 blue (AmpliSens, Россия), 1,4 мкл эквимолярной смеси четырех дезоксирибонуклеозидтрифосфатов со стоковой концентрацией 4,4 мМ, олигонуклеотидные последовательности, которые содержат индексы, например олигонуклеотиды для индексации Nextera [https://dnatech.genomecenter.ucdavis.edu/wp-content/uploads/2019/03/illumina-adapter-sequences-2019-1000000002694-10.pdf], в концентрации 0,2 пмоль/мкл и воду milli-Q. ДНК-матрица добавляется в количестве 5 мкл, итоговый объем реакции составляет 25 мкл. Температурный профиль индексации указан в Таблице 3. 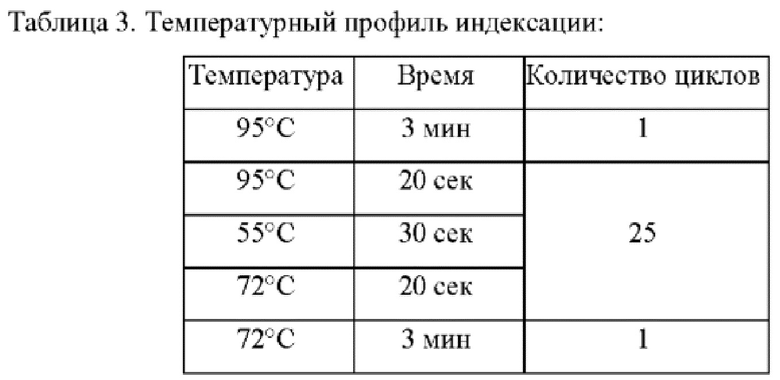
Олигонуклеотиды, используемые для индексации, в свою очередь, на одном конце содержат последовательности, соответствующие индексам, а на другом последовательности, комплементарные упомянутым ранее адаптерным последовательностям. Таким образом, в процессе индексации происходят этапы отжига олигонуклеотидов на концах целевых фрагментов и амплификации продукта.
Длины полученных продуктов амплификации могут быть проанализированы с помощью электрофореза в 1% агарозном геле. Далее повторно проводят очистку от реакционной смеси с использованием магнитных частиц, например, AMPure XP beads (Beckman Coulter, США) в объемном отношении 1:1.
В результате получаются готовые для секвенирования библиотеки, содержащие целевые фрагменты ДНК вируса гепатита В, которые позволяют определить полный геном ВГВ исходном образце ДНК.
Реализация заявляемого изобретения поясняется следующими примерами:
Пример 1. Получение набора олигонуклеотидных праймеров, используемого в способе пробоподготовки образцов для полногеномного секвенирования ДНК вируса гепатита В.
При разработке применяемых в заявляемом способе оригинальных синтезированных олигонуклеотидных праймеров для таргетной амплификации фрагментов вируса гепатита В, олигонуклеотидные последовательности были подобраны в полуавтоматическом режиме с учетом имеющейся информации о распространенных подтипах ВГВ.
Данный процесс включал в себя загрузку последовательностей геномов ВГВ из баз данных NCBI [https://www.ncbi.nlm.nih.gov/] и HBV db [https://hbvdb.lyon.inserm.fr], их выравнивание целью определения консервативных фрагментов и непосредственно генерацию праймерных последовательностей.
Температуры плавления полученных олигонуклеотидов определялись с помощью инструмента Multiple Primer Analyzer от Thermo Fisher Scientific (США). Подбор каждой новой праймерной пары в пуле осуществлялся таким образом, чтобы максимизировать сумму изменений термодинамических потенциалов Гиббса попарных взаимодействий всех последовательностей этого пула на данном шаге. Расчет термодинамических потенциалов проводился с помощью утилиты PrimerDimer. При этом учитывались дополнительные адаптерные части, добавляемые к праймерам на этапе синтеза, значительно удешевляющие и ускоряющие процесс индексации продуктов амплификации.
Затем, с помощью программы blastn оценивалась специфичность каждой полученной последовательности ко всем известным организмам, в частности Homo sapiens. Описанные действия позволили минимизировать неспецифическое взаимодействие между праймерами и участками ДНК человека и других организмов. Длины ампликонов были выбраны в пределах 300 п.н., что позволило избежать этапа отбора фрагментов с заданной длиной и добиться совместимости с большинством наборов реагентов для секвенирования от компании Illumina.
В результате были получены праймеры, имеющие структуры SEQ ID NO: 1-55. При этом олигонуклеотидные праймеры, имеющие структуры SEQ ID NO: 1, 3, 5, 8, 10, 12, 15, 16, 18, 22-24, 27, 28, 31, 34, 36, 38, 40, 42, 45, 47, 48, 51, 54 выполняют функции прямых праймеров, а олигонуклеотидные праймеры, имеющие структуры SEQ ID NO: 2, 4, 6, 7, 9, 11,13,14,17,19-21, 25, 26,29,30,32,33,35, 37, 39, 41, 43-44, 46, 49, 50, 52, 53, 55 выполняют функции обратных праймеров.
Кроме того, олигонуклеотидные праймеры содержат в своем составе универсальные адаптерные последовательности, прямые праймеры содержат адаптерную последовательность SEQ ID NO: 56, а обратные праймеры содержат адаптерную последовательность SEQ ID NO: 57. Данные последовательности позволяют в дальнейшем значительно оптимизировать процесс индексации продуктов амплификации.
Поскольку полученные олигонуклеотиды на одном конце имеют праймерную последовательность, комплементарную участкам генома вируса гепатита В, а на другом -адаптерные последовательности, следовательно, полученные после амплификации целевые фрагменты сразу были фланкированы такими адаптерными последовательностями.
Для индексации используются олигонуклеотиды, несущие различные индексы, которые позволяют отличать данные секвенирования молекул из разных образцов. Примеры подобных индексных последовательностей доступны на сайтах производителей наборов реагентов для высокопроизводительного секвенирования. Для реализации заявленного решения были использованы индексы Nextera [https://support-docs.illumina.com/SHARE/AdapterSeq/Content/SHARE/AdapterSeq/Nextera/DNAIndexesNXT.htm]. Структуры используемых для индексации олигонуклеотидов: 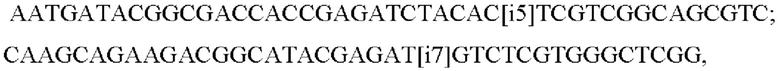
известны и используются для подготовки библиотек для секвенирования на платформе Illumina [https://dnatech.genomecenter.ucdavis.edu/wp-content/uploads/2019/03/illumina-adapter-sequences-2019-1000000002694-10.pdf]. Данные олигонуклеотиды на 3’-конце содержат последовательности, комплементарные адаптерным последовательностям SEQ ID NO: 56, 57.
Таким образом, в процессе индексации происходят этапы отжига олигонуклеотидов на концах целевых фрагментов и амплификации. После этого продукты амплификации подвергаются секвенированию методом NGS на платформе Illumina.
Пример 2. Полногеномное секвенирование вируса гепатита В в контрольном образце плазмы крови, подтип A2.
От научно-производственной лаборатории ФБУН ЦНИИ Эпидемиологии Роспотребнадзора был получен контрольный образец плазмы крови «КП ДНК HBV плазма №382916 с.10.10.08», содержащий ВГВ, вирусная нагрузка составила ~1,27*108 МЕ/мл. Выделение ДНК осуществлялось при помощи набора реагентов «РИБО-преп» (AmpliSens, Россия) согласно рекомендациям производителя.
Амплификация осуществлялась с применением оригинальных синтезированных олигонуклеотидных праймеров в двух реакциях, в каждой использовался отдельный пул олигонуклеотидов и выделенная ДНК в качестве матрицы.
Первый пул олигонуклеотидов содержал олигонуклеотиды SEQ ID NO: 1-30, второй пул олигонуклеотидов содержит олигонуклеотиды SEQ ID NO: 31-55. При этом, на концах прямых праймеров были расположены адаптерные последоватености SEQ ID NO: 56, на концах обратных праймеров - адаптерные последовательности SEQ ID NO: 57.
ПЦР-микс объемом 15 мкл на одну реакцию содержал 10 мкл ПЦР-смеси-2 blue (AmpliSens, Россия), 1,4 мкл эквимолярной смеси четырех дезоксирибонуклеозидтрифосфатов со стоковой концентрацией 4,4 мМ, заявляемые олигонуклеотиды в указанных в Таблице 1 концентрациях и воду milli-Q. ДНК-матрицу добавляли в количестве 10 мкл, итоговый объем реакции составлял 25 мкл. Температурный профиль амплификации для получения ПЦР-продукта указан в Таблице 4.
Таблица 4. Температурный профиль амплификации для получения ПЦР-продукта для полногеномного секвенирования вируса гепатита В в контрольном образце плазмы крови, подтип А2.
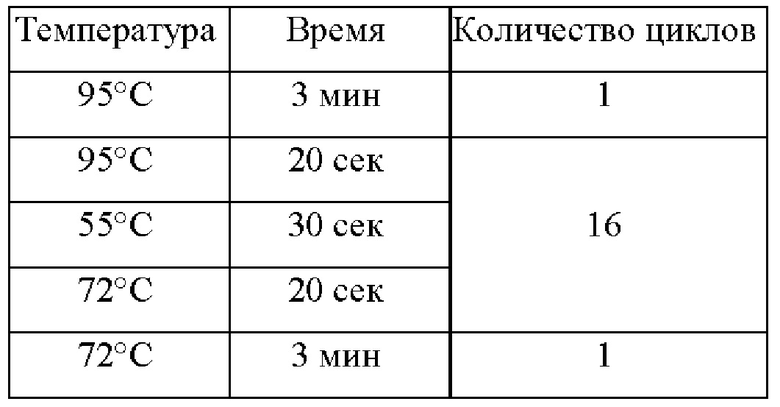
Далее проводили очистку ПЦР-продуктов от реакционной смеси с использованием магнитных частиц AMPure XP beads (Beckman Coulter, США) в объемном отношении 1:1.
ПЦР-микс объемом 20 мкл на одну реакцию содержал 10 мкл ГЩР-смеси-2 blue (AmpliSens, Россия), 1,4 мкл эквимолярной смеси четырех дезоксирибонуклеозидтрифосфатов со стоковой концентрацией 4,4 мМ, олигонуклеотидные последовательности для индексации, описанные в Примере 1, в концентрации 0,2 пмоль/мкл и воду milli-Q. ДНК-матрица добавлялась в количестве 5 мкл, итоговый объем реакции составлял 25 мкл. Температурный профиль индексации указан в Таблице 5.
Таблица 5. Температурный профиль индексации для получения ПЦР-продукта для полногеномного секвенирования вируса гепатита В в контрольном образце плазмы крови, подтип А2.
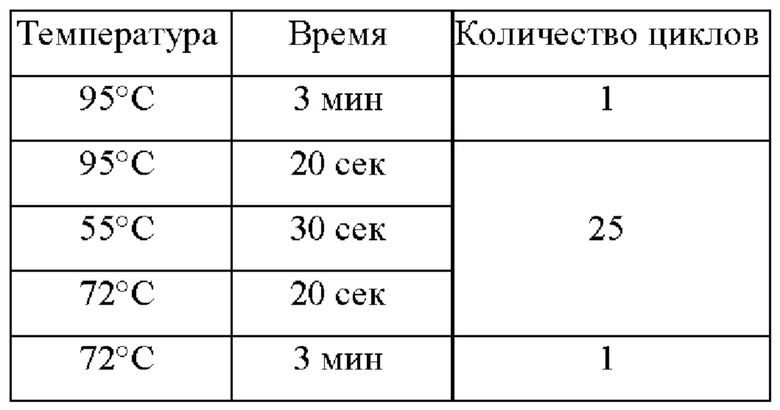
Далее повторно проводили очистку от реакционной смеси с использованием магнитных частиц AMPure XP beads (Beckman Coulter, США) в объемном отношении 1:1. В результате получились готовые для секвенирования библиотеки, содержащие целевые фрагменты ДНК вируса гепатита В, которые позволяют определить полный геном ВГВ в исходном образце ДНК.
Данные библиотеки были секвенированы на платформе Illumina с использованием набора реагентов для секвенирования MiSeq Reagent Kit v3 (600-cycle) Illumina (США). Результаты исследования были проанализированы биоинформатически с помощью утилит FastQC, bwa и samtools. По результатам биоинформатического анализа была определена полная нуклеотидная последовательность ДНК вируса гепатита В в исходном образце: 
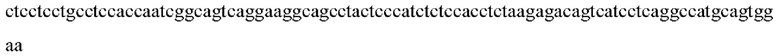
Данная последовательность была подтверждена секвенированием по методу Сэнгера. Из референсных геномов ВГВ данная последовательность оказалась наиболее близка геному Х02763, подтип А2 [https://www.ncbi.nlm.nih.gov/nuccore/X02763].
Пример 3. Полногеномное секвенирование вируса гепатита В в контрольном образце плазмы крови, подтип D1.
Из клинического образца плазмы крови «LA1», содержащего ВГВ, была выделена ДНК при помощи набора реагентов «РИБО-преп» (AmpliSens, Россия) согласно рекомендациям производителя. Вирусная нагрузка в образце составляла 1,95* 106 МЕ/мл.
Амплификация осуществлялась с применением оригинального синтезированного набора олигонуклеотидных праймеров в двух реакциях, в каждой использовался отдельный пул олигонуклеотидов и выделенная ДНК в качестве матрицы.
Первый пул олигонуклеотидов содержал олигонуклеотиды SEQ ID NO: 1-30, второй пул олигонуклеотидов содержит олигонуклеотиды SEQ ID NO: 31-55. При этом, на концах прямых праймеров расположены адаптерные последоватености SEQ ID NO: 56, на концах обратных праймеров расположены адаптерные последовательности SEQ ID NO: 57.
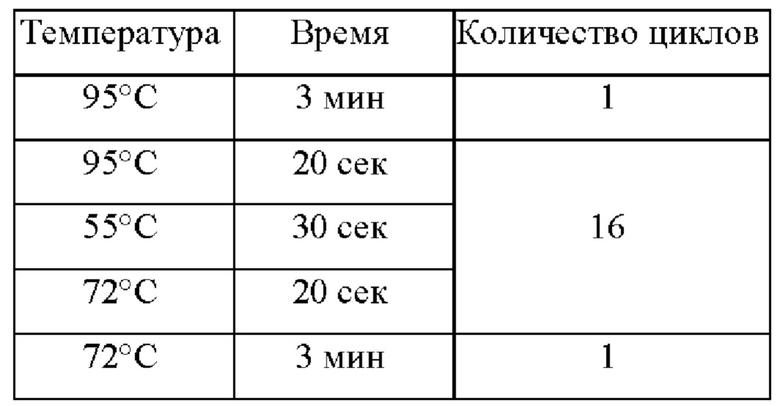
ПЦР-микс объемом 15 мкл на одну реакцию содержал 10 мкл ПЦР-смеси-2 blue (АмплиСенс, Россия), 1,4 мкл эквимолярной смеси четырех дезоксирибонуклеозидтрифосфатов со стоковой концентрацией 4,4 мМ, заявляемые олигонуклеотиды в указанных в Таблице 1 концентрациях и воду milli-Q. ДНК-матрицу добавляли в количестве 10 мкл, итоговый объем реакции составлял 25 мкл. Температурный профиль амплификации для получения ПЦР-продукта представлен в Таблице 6.
Таблица 6. Температурный профиль амплификации для получения ПЦР-продукта для полногеномного секвенирования вируса гепатита В в контрольном образце плазмы крови, подтип D1
Далее проводили очистку ПЦР-продуктов от реакционной смеси с использованием магнитных частиц AMPure XP beads (Beckman Coulter, США) в объемном отношении 1:1.
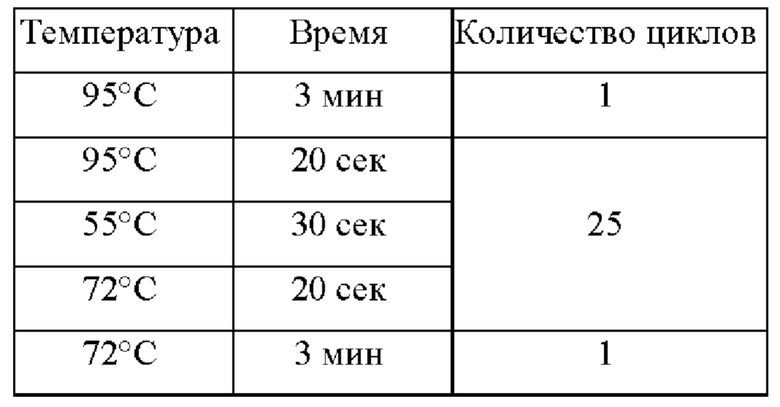
ПЦР-микс объемом 20 мкл на одну реакцию содержал 10 мкл ПЦР-смеси-2 blue (AmpliSens, Россия), 1,4 мкл эквимолярной смеси четырех дезоксирибонуклеозидтрифосфатов со стоковой концентрацией 4,4 мМ, олигонуклеотидные последовательности для индексации, описанные в Примере 1, в концентрации 0,2 пмоль/мкл и воду milli-Q. ДНК-матрица добавлялась в количестве 5 мкл, итоговый объем реакции составлял 25 мкл. Температурный профиль индексации указан в Таблице 7.
Таблица 7. для полногеномного секвенирования вируса гепатита В в контрольном образце плазмы крови, подтип D1.
Далее повторно проводили очистку от реакционной смеси с использованием магнитных частиц AMPure XP beads (Beckman Coulter, США) в объемном отношении 1:1. В результате получились готовые для секвенирования библиотеки, содержащие целевые фрагменты ДНК вируса гепатита В, которые позволяют определить полный геном ВГВ исходном образце ДНК.
Данные библиотеки были секвенированы на платформе Illumina с использованием набора реагентов для секвенирования MiSeq Reagent Kit v3 (600-cycle) Illumina (США). Результаты исследования были проанализированы биоинформатически с помощью утилит FastQC, bwa и samtools. По результатам биоинформатического анализа была определена полная нуклеотидная последовательность ДНК вируса гепатита В в исходном образце:


Данная последовательность была подтверждена секвенированием по Сэнгеру. Из референсных геномов ВГВ данная последовательность оказалась наиболее близка геному AF 121240, подтип D1 [https://www.ncbi.nlm.nih.gov/nuccore/AF121240].
Пример 4. Полногеномное секвенирование вируса гепатита В в контрольном образце плазмы крови, подтип В2.
Из клинического образца плазмы крови «LF100», содержащего вирус гепатита В, была выделена ДНК при помощи набора реагентов «РИБО-преп» (AmpliSens, Россия) согласно рекомендациям производителя, вирусная нагрузка в образце составляла 2,62* 107 МЕ/мл.
Амплификация осуществлялась с применением оригинального синтезированного набора олигонуклеотидных праймеров в двух реакциях, в каждой использовался отдельный пул олигонуклеотидов и выделенная ДНК в качестве матрицы.
Первый пул олигонуклеотидов содержал олигонуклеотиды SEQ ID NO: 1-30, второй пул олигонуклеотидов содержит олигонуклеотиды SEQ ID NO: 31-55. При этом, на концах прямых праймеров расположены адаптерные последоватености SEQ ID NO: 56, на концах обратных праймеров расположены адаптерные последовательности SEQ ID NO: 57.
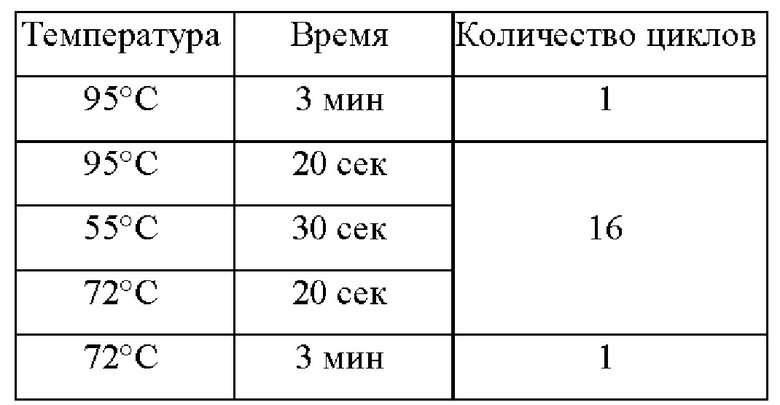
ПЦР-микс объемом 15 мкл на одну реакцию содержал 10 мкл ПЦР-смеси-2 blue (AmpliSens, Россия), 1,4 мкл эквимолярной смеси четырех дезоксирибонуклеозидтрифосфатов со стоковой концентрацией 4,4 мМ, заявляемые олигонуклеотиды в указанных в Таблице 1 концентрациях и воду milli-Q. ДНК-матрицу добавляли в количестве 10 мкл, итоговый объем реакции составлял 25 мкл. Температурный профиль амплификации для получения ПЦР-продукта представлен в Таблице 8.
Таблица 8. Температурный профиль амплификации для получения ПЦР-продукта для полногеномного секвенирования вируса гепатита В в контрольном образце плазмы крови, подтип В2
Далее проводили очистку ПЦР-продуктов от реакционной смеси с использованием магнитных частиц AMPure XP beads (Beckman Coulter, США) в объемном отношении 1:1.
ПЦР-микс объемом 20 мкл на одну реакцию содержал 10 мкл ПЦР-смеси-2 blue (AmpliSens, Россия), 1,4 мкл эквимолярной смеси четырех дезоксирибонуклеозидтрифосфатов со стоковой концентрацией 4,4 мМ, олигонуклеотидные последовательности для индексации, описанные в Примере 1, в концентрации 0,2 пмоль/мкл и воду milli-Q. ДНК-матрица добавлялась в количестве 5 мкл, итоговый объем реакции составлял 25 мкл. Температурный профиль индексации представен в Таблице 9.
Таблица 9. Температурный профиль индексации для полногеномного секвенирования вируса гепатита В в контрольном образце плазмы крови, подтип В2.
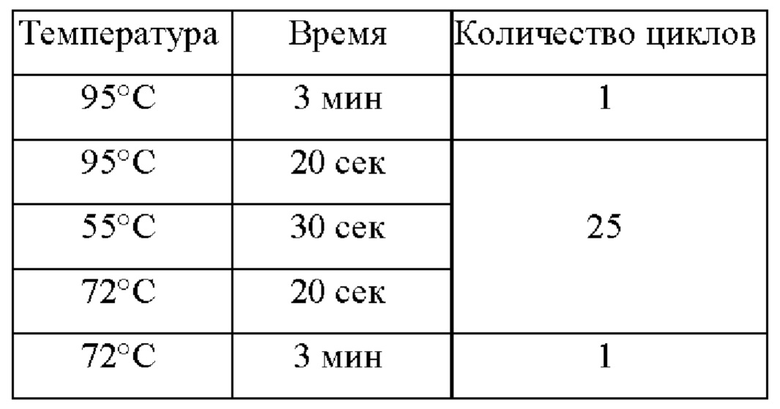
Далее повторно проводили очистку от реакционной смеси с использованием магнитных частиц AMPure XP beads (Beckman Coulter, США) в объемном отношении 1:1. В результате получились готовые для секвенирования библиотеки, содержащие целевые фрагменты ДНК вируса гепатита В, которые позволяют определить полный геном ВГВ исходном образце ДНК.
Данные библиотеки были секвенированы на платформе Illumina с использованием набора реагентов для секвенирования MiSeq Reagent Kit v3 (600-cycle) Illumina (США). Результаты исследования были проанализированы биоинформатически с помощью утилит FastQC, bwa и samtools. По результатам биоинформатического анализа была определена полная нуклеотидная последовательность ДНК вируса гепатита В в исходном образце:


Данная последовательность была подтверждена секвенированием по методу Сэнгера. Из референсных геномов ВГВ данная последовательность оказалась наиболее близка геному АВ 219428, подтип В2 [https://www.ncbi.nlm.nih.gov/nuccore/AB219428].
Таким образом, заявленное решение позволяет произвести пробоподготовку библиотек, содержащие целевые фрагменты генома вируса гепатита В, с наименьшими трудовыми, временными и материальными затратами, не снижая качество и объемы произведенных исследований
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<ST26SequenceListing dtdVersion="V1_2" fileName="Oligonukleotidy dlya
probopodgotovki obrazcov DNK virusa gepatita V" softwareName="WIPO
Sequence"
softwareVersion="1.0.0" productionDate="2023-08-24">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>No</ApplicationNumberText>
<FilingDate>2023-08-24</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>1</ApplicantFileReference>
<ApplicantName languageCode="ru">ФБУН ЦНИИ Эпидемиологии
Роспотребнадзора</ApplicantName>
<ApplicantNameLatin>FBUN CRIE</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ пробоподготовки образцов ДНК
вируса гепатита В для полногеномного секвенирования и
олигонуклеотидные праймеры для его реализации</InventionTitle>
<SequenceTotalQuantity>57</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctggtggctccagttcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaggacaacaggttggtgag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcccgtttgtcctctaattc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccagacagtgggggaaag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cacaaaacaaaaagatgggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaaaggccttgtaagttggcga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaaaggccttatacgttggcga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctntgccaagtgtttgctga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcgmaggatccagttggc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gggcgcacctctctttac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aagtatgcctcaaggtcgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggaggctgtaggcataaattg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caaaaacgagagtaactccacag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggcaaaaaagagaacaattccacag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctctagcyacctgggtggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atctagcyacctgggtggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgcgaggcgagggagttc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caatcgccgcgtcgcaga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cccttatccaatggtaaatatttngt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tttctctaggggcaaatatttngt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gatacctttgtctaatggtaaatatttngt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctatttacacactctatggaaggc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="23">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cattatttacatactctttggaaggc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="24">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atttacacaccctatggaaggc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="25">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttcgggaaagaatcccagagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="26">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgggaacacatcccagagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="27">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgcaaatccagattgggac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="28">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agcagtcccgactgggac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="29">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atcttgcagagtttggtggaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="30">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atctagcagagcttgatgaaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="31">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cacagagtctagactcgtggtg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="32">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcaggttctgcatggtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="33">
<INSDSeq>
<INSDSeq_length>16</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aggttcggcagggtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="34">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccatttgttcagtggttcgtag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="35">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atcaataggcctgtttacaggaag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="36">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaaagtatgtcaaagaattgtggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="37">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cagtatggatcggcagagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="38">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcctagccgcttgttttg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="39">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtccggcagatgagaagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="40">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcatggagaccaccgtgaac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="41">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcacagcttggaggcttgaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="42">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>attgacccgtataaagaatttgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="43">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcctgaactttaggcccatattagt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="44">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcctgatttttaaacccatattagt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="45">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cggagtgtggattcgcac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="46">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cccaccttatgagtccaagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="47">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaaagaagattgcaattgattatgcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="48">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaagaagattaaacttagtcatgcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="49">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agatcttgttcccaagaatatgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="50">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtgttcccgagaataagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="51">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggaacaagatctacagcatggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="52">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tattggtggaggcaggagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="53">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgattggtagaagcaggagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="54">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctcaggccatgcagtgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="55">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aacacgagmaggggtcctag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="56">
<INSDSeq>
<INSDSeq_length>33</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..33</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="57">
<INSDSeq>
<INSDSeq_length>34</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>HBV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctcgtgggctcggagatgtgtataagagacag</INSDSeq_sequence
>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
Изобретение относится к биотехнологии, медицине, молекулярной биологии. Предложен способ пробоподготовки образцов ДНК, включающий стадии: (а) выделения ДНК из биологического материала; (b) проведения ПЦР с применением двух пулов олигонуклеотидных праймеров, где для первого пула применяют олигонуклеотидные праймеры, имеющие структуру SEQ ID NO: 1-30, а для второго пула применяют олигонуклеотидные праймеры, имеющие структуру SEQ ID NO: 31-55; (с) очистки продуктов ПЦР при помощи магнитных частиц; (d) индексации полученных ампликонов при помощи праймеров, содержащих адаптерные последовательности SEQ ID NO: 56, 57; (е) повторной очистки продуктов ПЦР при помощи магнитных частиц; (f) оценки получаемых в ходе амплификации ПЦР-продуктов с помощью электрофореза. Предложен пул олигонуклеотидных праймеров для реализации способа, имеющих структуру SEQ ID NO: 1-57, для выполнения функции праймеров, комплементарных участкам гена вируса гепатита В. Изобретение позволяет произвести пробоподготовку библиотек, содержащих целевые фрагменты генов вируса гепатита В (ВГВ), методом высокопроизводительного секвенирования для целей поиска мутаций вируса гепатита В, не снижая качество и объемы произведенных исследований. 3 н. и 7 з.п. ф-лы, 9 табл., 4 пр.
1. Способ пробоподготовки образцов ДНК вируса гепатита В для полногеномного секвенирования, включающий стадии:
(a) выделения ДНК из биологического материала;
(b) проведения ПЦР с применением двух пулов олигонуклеотидных праймеров, где для первого пула применяют олигонуклеотидные праймеры, имеющие структуру SEQ ID NO: 1-30, а для второго пула применяют олигонуклеотидные праймеры, имеющие структуру SEQ ID NO: 31-55;
(c) очистки продуктов ПЦР при помощи магнитных частиц;
(d) индексации полученных ампликонов при помощи праймеров, содержащих адаптерные последовательности SEQ ID NO: 56, 57;
(e) повторной очистки продуктов ПЦР при помощи магнитных частиц;
(f) оценки получаемых в ходе амплификации ПЦР-продуктов с помощью электрофореза.
2. Способ по п. 1, где реакционная смесь для первого пула содержит олигонуклеотидные праймеры в следующей концентрации: SEQ ID NO: 3, 4, 10, 11, 15, 16, 17 - по 0,05 пмоль/мкл каждый; SEQ ID NO: 18, 19, 20, 21 - по 0,2 пмоль/мкл каждый; SEQ ID NO: 1, 2, 5-9, 12, 14, 22-30 - по 0,1 пмоль/мкл каждый.
3. Способ по п. 1, где реакционная смесь для второго пула содержит олигонуклеотидные праймеры в следующей концентрации: SEQ ID NO: 51-55 - по 0,05 пмоль/мкл каждый; SEQ ID NO: 38, 39, 42-44, 47-50 - по 0,2 пмоль/мкл каждый; SEQ ID NO: 31-37, 40, 41, 45, 46 - по 0,1 пмоль/мкл каждый.
4. Пул олигонуклеотидных праймеров для реализации способа по п. 1, имеющих структуру SEQ ID NO: 1-30, для выполнения функции праймеров, комплементарных участкам генов вируса гепатита В.
5. Пул олигонуклеотидных праймеров по п. 4, где функции прямых праймеров выполняют олигонуклеотидные праймеры, имеющие структуру SEQ ID NO: 1, 3, 5, 8, 10, 12, 15, 16, 18, 22-24, 27, 28.
6. Пул олигонуклеотидных праймеров по п. 4, где функции обратных праймеров выполняют олигонуклеотидные праймеры, имеющие структуру SEQ ID NO: 2, 4, 6, 7, 9, 11, 13, 14, 17, 19-21, 25, 26, 29, 30.
7. Пул олигонуклеотидных праймеров для реализации способа по п. 1, имеющих структуру SEQ ID NO: 31-55, для выполнения функции праймеров, комплементарных участкам генов вируса гепатита В.
8. Пул олигонуклеотидных праймеров по п. 7, где функции прямых праймеров выполняют олигонуклеотидные праймеры, имеющие структуру SEQ ID NO: 31, 34, 36, 38, 40, 42, 45, 47, 48, 51, 54.
9. Пул олигонуклеотидных праймеров по п. 7, где функции обратных праймеров выполняют олигонуклеотидные праймеры, имеющие структуру SEQ ID NO: 32, 33, 35, 37, 39, 41, 43-44, 46, 49, 50, 52, 53, 55.
10. Пул олигонуклеотидных праймеров по пп. 4 и 7, где олигонуклеотиды в своем составе имеют адаптерные последовательности, при этом адаптерные последовательности SEQ ID NO: 56 расположены на концах олигонуклеотидных праймеров, имеющих структуру SEQ ID NO: 1, 3, 5, 8, 10, 12, 15, 16, 18, 22-24, 27, 28, 31, 34, 36, 38, 40, 42, 45, 47, 48, 51, 54, а адаптерные последовательности SEQ ID NO: 57 расположены на концах олигонуклеотидных праймеров, имеющих структуру SEQ ID NO: 2, 4, 6, 7, 9, 11, 13, 14, 17, 19-21, 25, 26, 29, 30, 32, 33, 35, 37, 39, 41, 43-44, 46, 49, 50, 52, 53, 55.
| ГЕНОТИПИРОВАНИЕ DEEPCHEK®-HBV RT И АНАЛИЗ DR, опубликовано согласно Wayback Machine 27.11.2020, весь документ, https://www.ablsa.com/laboratory-applications/deepchek-hbv-rt-genotyping-dr-assay | |||
| EA 18102 B1, 30.05.2013 | |||
| Способ обнаружения ДНК вируса гепатита B в ультранизких концентрациях и специфические олигонуклеотиды для использования в способе | 2021 |
|
RU2782951C1 |
| Penelope Garmiri et al., Full Genome Characterization of Hepatitis B Virus Strains From Blood Donors | |||
Авторы
Даты
2024-05-03—Публикация
2023-09-11—Подача