Область техники
[0001] Настоящее изобретение относится к способу получения линейной или кольцевой ДНК посредством связывания фрагментов ДНК двух или более типов друг с другом в областях, имеющих гомологичные последовательности оснований, и к набору для связывания фрагментов ДНК, используемому в этом способе.
В настоящей заявке испрашивается приоритет заявки на патент Японии № 2017-132084, поданной 5 июля 2017, и заявки на патент Японии № 2017-231732, поданной 1 декабря 2017, содержание которых включено в настоящее описание посредством ссылки.
Уровень техники
[0002] В настоящей заявке описан способ получения линейной или кольцевой двухцепочечной ДНК посредством связывания множества линейных двухцепочечных фрагментов ДНК. В этом способе может быть получена более длинная двухцепочечная ДНК, которую трудно синтезировать путем химического синтеза. Способы связывания линейных двухцепочечных фрагментов ДНК, главным образом, включают способ лигирования (Патентный документ 1) и способ сборки Гибсона (Патентный документ 2 и Патентный документ 3).
[0003] Способ лигирования представляет собой способ, в котором реакцию связывания осуществляют с использованием гибридного фермента, обладающего функцией распознавания гомологичной последовательности концевых 15 оснований каждого двухцепочечного фрагмента ДНК и их связывания. В частности, сначала, гомологичную область, состоящую из последовательности одинаковых оснований, присоединяют к концам двух двухцепочечных фрагментов ДНК, связываемых с помощью ПЦР. Два двухцепочечных фрагмента ДНК, по обоим концам которых была присоединена гомологичная область из 15 оснований, связывают путем смешивания с гибридным ферментом и инкубирования.
[0004] С другой стороны, в способе сборки Гибсона, сначала дистальную область первой молекулы ДНК и проксимальную область второй молекулы ДНК расщепляют с использованием фермента, обладающего экзонуклеазной активностью. В результате, соответствующие гомологичные области (области с идентичными последовательностями, которые являются достаточно длинными для специфической гибридизации друг с другом) становятся одноцепочечными. Затем, после их специфического отжига и связывания, гэпы и ники подвергают репарации с получением полноразмерной молекулы со связанными двухцепочечными ДНК. Так, например, в Патентном документе 2 раскрываются двухцепочечные фрагменты ДНК размером 1,6 т.п.о. и 1,4 т.п.о., полученные в одноцепочечной форме путем расщепления ДНК-полимеразой Т4, обладающей экзонуклеазной активностью. Полученные две одноцепочечные ДНК были связаны друг с другом в присутствии добавленных RecA, dNTP и ДНК-лигазы, а затем гэп был достроен под действием полимеразной активности этой ДНК-полимеразы Т4 для репарации ников, и была получена двухцепочечная ДНК размером 3 т.п.о.
[0005] Кроме того, в патентном документе 4 описан способ связывания множества линейных двухцепочечных фрагментов ДНК с использованием RecA. Этот способ включает стадию инкубирования множества линейных двухцепочечных фрагментов ДНК с ДНК-лигазой в присутствии рекомбиназы семейства RecA или белка, обладающего рекомбинантной активностью, с получением линейной двухцепочечной ДНК, в которой множество линейных двухцепочечных фрагментов ДНК связаны друг с другом последовательно. В этом способе, с использованием ферментов-рекомбиназ семейства RecA или т.п. происходит ингибирование связывания обоих концов линейного двухцепочечного фрагмента ДНК и стимуляция связывания концов линейных двухцепочечных фрагментов ДНК.
Список цитируемых документов
Патентный документ
[0006] Патентный документ 1:. Патент США № 7575860.
Патентный документ 2: Патент США № 7776532.
Патентный документ 3: Патент США № 8968999.
Патентный документ 4: Нерассмотренная заявка на патент Японии, первая публикация № 2016-077180.
Сущность изобретения
Техническая проблема
[0007] С применением способа лигирования и способа сборки Гибсона, два линейных фрагмента ДНК могут быть связаны друг с другом без каких-либо проблем. Однако недостаток этих способов заключается в том, что число связанных фрагментов увеличивается, а эффективность связывания снижается, а поэтому не может быть получена представляющая интерес связанная молекула.
[0008] Целью настоящего изобретения является разработка способа получения линейной или кольцевой ДНК посредством связывания фрагментов ДНК двух или более типов друг с другом в областях, имеющих гомологичные последовательности оснований, и получение набора для связывания фрагментов ДНК, используемого в этом способе.
Средства для решения данной проблемы
[0009] В результате проведения интенсивных исследований, авторами настоящего изобретения было обнаружено, что ДНК двух или более типов могут быть эффективно связаны с использованием рекомбиназы семейства RecA и экзонуклеазы, и на этой основе было создано настоящее изобретение.
[0010] Способ получения ДНК и набор для связывания фрагментов ДНК согласно изобретению представлены ниже в [1]-[31].
[1] Способ получения ДНК, где указанный способ включает:
получение реакционного раствора, содержащего фрагменты ДНК двух или более типов и белок, обладающий рекомбиназной активностью семейства RecA, и
получение линейной или кольцевой ДНК в реакционном растворе путем связывания фрагментов ДНК двух или более типов друг с другом в областях, имеющих гомологичные последовательности оснований или в областях, имеющих комплементарные последовательности оснований.
[2] Способ получения ДНК в соответствии с [1], где реакционный раствор также содержит экзонуклеазу.
[3] Способ получения ДНК в соответствии с [2], где экзонуклеазой является 3'→5'-экзонуклеаза.
[4] Способ получения ДНК в соответствии с [1], где реакционный раствор также содержит 3'→5'-экзонуклеазу, специфичную к линейной двухцепочечной ДНК.
[5] Способ получения ДНК в соответствии с [1], где реакционный раствор также содержит 3'→5'-экзонуклеазу, специфичную к линейной двухцепочечной ДНК, и 3'→5'-экзонуклеазу, специфичную к одноцепочечной ДНК.
[6] Способ получения ДНК в соответствии с любым из [1]- [5], где реакционный раствор содержит регенерирующий фермент для нуклеозид-трифосфатов или дезоксинуклеотид-трифосфатов и его субстрат.
[7] Способ получения ДНК в соответствии с [6], где регенерирующим ферментом является креатинкиназа, а его субстратом является фосфат креатина; регенерирующим ферментом является пируват-киназа, а его субстратом является пируват фосфоэнола; регенерирующим ферментом является ацетат-киназа, а его субстратом является ацетилфосфат; регенерирующим ферментом является полифосфат-киназа, а его субстратом является полифосфат; или регенерирующим ферментом является нуклеозид-дифосфат-киназа, а его субстратом является нуклеозид-трифосфат.
[8] Способ получения ДНК в соответствии с любым из [1]-[7], где реакционный раствор в начале реакции связывания фрагментов ДНК двух или более типов имеет концентрацию источника ионов магния 0,5-15 мМ, и концентрацию нуклеозид-трифосфата или дезоксинуклеотид-трифосфата 1-1000 мкМ.
[9] Способ получения ДНК в соответствии с любым из [1]-[8], где реакцию связывания фрагментов ДНК двух или более типов осуществляют при температуре в пределах 25-48°C.
[10] Способ получения ДНК в соответствии с любым из [1]-[9], где линейную или кольцевую ДНК получают путем связывания 7 или более фрагментов ДНК.
[11] Способ получения ДНК в соответствии с любым из [1]-[10], где реакционный раствор содержит одно или более веществ, выбранных из группы, состоящей из хлорида тетраметиламмония и диметилсульфоксида.
[12] Способ получения ДНК в соответствии с любым из [1]-[11], где реакционный раствор содержит одно или более веществ, выбранных из группы, состоящей из полиэтиленгликоля, источника ионов щелочного металла и дитиотреитола.
[13] Способ получения ДНК в соответствии с любым из [1]-[12], где белком, обладающим рекомбиназной активностью семейства RecA, является uvsX, и реакционный раствор также содержит uvsY.
[14] Способ получения ДНК в соответствии с любым из [1]-[13], где области, имеющие гомологичные последовательности оснований, или области, имеющие комплементарные последовательности оснований, присутствуют на конце или возле конца фрагмента ДНК.
[15] Способ получения ДНК в соответствии с [14], где области, имеющие гомологичные последовательности оснований, или области, имеющие комплементарные последовательности оснований, имеют длину от 10 п.о. до 500 п.о.
[16] Способ получения ДНК в соответствии с любым из [1]-[15], где реакционный раствор в начале реакции связывания фрагментов ДНК двух или более типов содержит фрагменты ДНК двух или более типов в одной и той же молярной концентрации.
[17] Способ получения ДНК в соответствии с любым из [1]-[16], также включающий репарацию гэпов и ников в полученной линейной или кольцевой ДНК с использованием ферментов для репарации гэпов.
[18] Способ получения ДНК в соответствии с [17], также включающий термообработку полученной линейной или кольцевой ДНК при 50-70°C с последующим быстрым охлаждением до температуры 10°C или менее, а затем репарацией гэпов и ников с использованием ферментов для репарации гэпов.
[19] Способ получения ДНК в соответствии с [17] или [18], также включающий амплификацию линейной или кольцевой двухцепочечной ДНК с репарацией гэпов и ников.
[20] Способ получения ДНК в соответствии с любым из [1]-[16], где ДНК, полученная путем связывания, является линейной, а ПЦР проводят непосредственно с использованием линейной ДНК в качестве матрицы.
[21] Способ получения ДНК в соответствии с любым из [1]-[16], где ДНК, полученная путем связывания, является кольцевой ДНК, содержащей последовательность ориджина репликации, способную связываться с ферментом, обладающим активностью DnaA, и
образовывать реакционную смесь, содержащую кольцевую ДНК, первую группу ферментов, которые катализируют репликацию кольцевой ДНК; вторую группу ферментов, которые катализирует реакцию связывания фрагментов Оказаки и синтезируют две сестринских кольцевых ДНК, состоящих из катенана; третью группу ферментов, которые катализируют разделение двух сестринских кольцевых ДНК, и dNTP.
[22] Способ получения ДНК в соответствии с [21], также включающий предварительную термообработку полученной ДНК при 50-70°С, с последующим быстрым охлаждением до температуры 10°C или менее, а затем получением реакционной смеси.
[23] Способ получения ДНК в соответствии с любым из [1]-[16], также включающий введение полученной линейной или кольцевой ДНК в микроорганизм и амплификацию двухцепочечной ДНК с репарацией гэпов и ников.
[24] Набор для связывания фрагментов ДНК, содержащий: белок, обладающий рекомбиназной активностью семейства RecA, где указанный набор используют для получения линейной или кольцевой ДНК посредством связывания фрагментов ДНК двух или более типов друг с другом в областях, имеющих гомологичные последовательности оснований, или в областях, имеющих комплементарные последовательности оснований.
[25] Набор для связывания фрагментов ДНК в соответствии с [24], также включающий экзонуклеазу.
[26] Набор для связывания фрагментов ДНК в соответствии с [25], где экзонуклеазой является 3'→5'-экзонуклеаза.
[27] Набор для связывания фрагментов ДНК в соответствии с [24], также содержащий экзонуклеазу, специфичную к линейной двухцепочечной ДНК.
[28] Набор для связывания фрагментов ДНК в соответствии с [24], также содержащий экзонуклеазу, специфичную к линейной двухцепочечной ДНК, и 3'→5'-экзонуклеазу, специфичную к одноцепочечной ДНК.
[29] Набор для связывания фрагментов ДНК в соответствии с любым из [24]-[28], который также содержит регенерирующий фермент для нуклеозид-трифосфатов или дезоксинуклеотид-трифосфатов и его субстрат.
[30] Набор для связывания фрагментов ДНК в соответствии с любым из [24]-[29], также содержащий одно или более веществ, выбранных из группы, состоящей из хлорида тетраметиламмония и диметилсульфоксида.
[31] Набор для связывания фрагментов ДНК в соответствии с любым из [24]-[30], также содержащий одно или более веществ, выбранных из группы, состоящей из нуклеозид-трифосфата, дезоксинуклеотид-трифосфата, источника ионов магния, источника ионов щелочного металла, полиэтиленгликоля, дитиотреитола и буфера.
Преимущественные эффекты изобретения
[0011] С применением способа получения ДНК согласно изобретению может быть осуществлено эффективное связывание множества фрагментов ДНК, в результате чего может быть получена линейная или кольцевая ДНК.
С помощью набора для связывания ДНК, способ получения ДНК может быть упрощен, и фрагменты ДНК могут быть эффективно связаны друг с другом.
Краткое описание чертежей
[0012] На фиг. 1 представлена диаграмма, схематически иллюстрирующая вариант, в котором линейные двухцепочечные фрагменты ДНК связывают друг с другом в соответствии с принципом осуществления способа получения ДНК согласно изобретению.
На фиг. 2 представлено цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционного раствора, полученного посредством реакции связывания 7 фрагментов в Примере 1.
На фиг. 3 представлено цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционного раствора, полученного посредством реакции связывания 5 фрагментов в Примере 2.
На фиг. 4 представлено цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционного раствора, полученного посредством реакции связывания 5 фрагментов в Примере 3.
На фиг. 5 представлено цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционного раствора, полученного посредством реакции связывания 7 фрагментов в Примере 4.
На фиг. 6 представлено цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционного раствора, полученного посредством реакции связывания 5 фрагментов в Примере 5.
На фиг. 7 представлено цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционного раствора, полученного посредством реакции связывания 7 фрагментов в Примере 5.
На фиг. 8 представлено цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционного раствора, полученного посредством реакции связывания 7 фрагментов в Примере 6.
На фиг. 9 представлено цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционного раствора, полученного посредством реакции связывания 7 фрагментов в Примере 7.
На фиг. 10 представлено цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционного раствора, полученного посредством реакции связывания 20-49 фрагментов в Примере 8.
На фиг. 11 представлено цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционного раствора, полученного посредством реакции связывания 25 фрагментов в Примере 9.
На фиг. 12 представлено (а) цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционного раствора, полученного посредством реакции связывания 21 или 26 фрагментов, и (b) цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционной смеси, полученной путем дополнительной RCR-амплификации после реакции связывания в Примере 10.
На фиг. 13 представлено (а) цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционного раствора, полученного посредством реакции связывания 26 фрагментов, и (b) цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционной смеси, полученной путем дополнительной RCR-амплификации после реакции связывания в Примере 11.
На фиг. 14 представлено (а) цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционного раствора, полученного посредством реакции связывания 26 или 36 фрагментов, и (b) цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционной смеси, полученной путем дополнительной RCR-амплификации после реакции связывания в Примере 12.
На фиг. 15 представлено (а) цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционного раствора, полученного посредством реакции связывания 26 фрагментов способом связывания (RA) согласно изобретению и способом NEB, и (b) цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционной смеси, полученной путем дополнительной RCR-амплификации после реакции связывания в Примере 13.
На фиг. 16 представлено цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционной смеси, полученной путем связывания Xba I-гидролизата геномной ДНК E. coli с фрагментом, содержащим oriC, с получением кольцевой ДНК с последующим проведением RCR-амплификации в Примере 14.
На фиг. 17 представлено цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционного раствора, полученного путем реакции связывания 10 фрагментов в реакционном растворе, содержащем систему регенерации АТФ, состоящую из креатин-киназы (СК) и фосфата креатина (СР) в Примере 15.
На фиг. 18 представлено цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционного раствора, полученного путем реакции связывания 10 фрагментов в реакционном растворе, содержащем систему регенерации АТФ, состоящую из креатин-киназы и фосфата креатина в Примере 16.
На фиг. 19 представлено цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционного раствора, полученного путем реакции связывания 36 фрагментов в реакционном растворе, содержащем систему регенерации АТФ, состоящую из пируват-киназы (PK) и пирувата фосфоэнола (РЕР) в Примере 17.
На фиг. 20 представлено цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционного раствора, полученного путем реакции связывания 10 фрагментов в реакционном растворе, содержащем систему регенерации АТФ, состоящую из полифосфат-киназы (PPK) и полифосфата (РР) в Примере 18.
На фиг. 21 представлено цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционного раствора, полученного путем реакции связывания 10 фрагментов в реакционном растворе, содержащем экзонуклеазу III и экзонуклеазу I в Примере 19.
На фиг. 22 представлено (а) цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционного раствора, полученного посредством реакции связывания 36 фрагментов и (b) цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционной смеси, полученной путем дополнительной RCR-амплификации после реакции связывания в Примере 20.
На фиг. 23 представлено (а) цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционного раствора, полученного посредством реакции связывания 50 или 36 фрагментов и (b) цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционной смеси, полученной путем дополнительной RCR-амплификации после реакции связывания в Примере 21.
На фиг. 24 представлено цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для ДНК, экстрагированной из трансформанта в Примере 21. Трансформант был получен путем введения ДНК в E. coli в реакционном растворе, полученном путем дополнительной RCR-амплификации после реакции связывания 50 фрагментов.
На фиг. 25 представлено цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для ферментативного расщепления амплифицированного продукта, полученного путем RCR-амплификации ДНК, экстрагированной из полученного трансформанта в Примере 21.
На фиг. 26 представлено цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционного раствора, полученного посредством реакции связывания 2 фрагментов в Примере 22.
На фиг. 27 представлено цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционного раствора, полученного путем реакции связывания 10 фрагментов в реакционном растворе, содержащем экзонуклеазу III, экзонуклеазу I и экзонуклеазу Т в Примере 23.
На фиг. 28 представлено цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционного раствора, полученного путем реакции связывания 10 фрагментов в реакционном растворе, содержащем UvsX, гомолог бактериофага RecA в Примере 24.
На фиг. 29 представлено цветное изображение полос, разделенных с помощью электрофореза в агарозном геле для реакционного раствора, полученного путем реакции связывания 10 фрагментов в реакционном растворе, содержащем UvsX и UvsY, в Примере 25.
Описание вариантов осуществления изобретения
[0013] <Способ получения ДНК>
В способе получения ДНК согласно изобретению, фрагменты ДНК с областями, имеющими последовательности оснований, гомологичные друг другу (далее иногда называемые просто «гомологичными областями»), или с областями, имеющими последовательности оснований, комплементарные друг другу (далее иногда называемые просто «комплементарными областями»), связывают друг с другом в гомологичных областях или в комплементарных областях с получением линейной или кольцевой ДНК. Поскольку способ получения ДНК согласно изобретению осуществляют посредством реакции связывания в присутствии белка, обладающего рекомбиназной активностью семейства RecA, то эффективность связывания этим способом является превосходной.
[0014] В настоящем изобретении и в настоящем описании, фраза «последовательности оснований являются гомологичными» означает, что «последовательности основания являются идентичными», а фраза «нуклеотидные последовательности являются комплементарными», означает, что «последовательности оснований комплементарны друг другу».
[0015] В частности, в способе получения ДНК согласно изобретению приготавливают реакционный раствор, содержащий фрагменты ДНК двух или более типов, и белок, обладающий рекомбиназной активностью семейства RecA (далее иногда называемый «рекомбиназным белком семейства RecA»), и фрагменты ДНК двух или более типов связывают друг с другом в гомологичных областях или в комплементарных областях в реакционном растворе. Этот способ позволяет получить линейную или кольцевую ДНК. Далее, линейная или кольцевая ДНК, в которой фрагменты ДНК двух или более типов являются связанными, может называться «связанной молекулой».
[0016] В способе получения ДНК согласно изобретению, связанный фрагмент ДНК может представлять собой линейный двухцепочечный фрагмент ДНК или одноцепочечный фрагмент ДНК. То есть, линейные двухцепочечные фрагменты ДНК могут быть связаны друг с другом; линейный двухцепочечный фрагмент ДНК и одноцепочечный фрагмент ДНК могут быть связаны друг с другом; или одноцепочечные фрагменты ДНК могут быть связаны друг с другом. Линейные двухцепочечные фрагменты ДНК одного или более типов и одноцепочечные фрагменты ДНК одного или более типов могут быть связанными. При связывании линейных двухцепочечных фрагментов ДНК или при связывании линейного двухцепочечного фрагмента ДНК и одноцепочечного фрагмента ДНК, оба этих фрагмента связывают друг с другом в гомологичной области. При связывании линейных одноцепочечных фрагментов ДНК, оба этих фрагмента связывают друг с другом в комплементарной области.
[0017] Если связанный фрагмент ДНК по меньшей мере одного типа представляет собой линейный двухцепочечный фрагмент ДНК в способе получения ДНК согласно настоящему изобретению, то реакционный раствор также содержит экзонуклеазу.
[0018] На фиг. 1 представлена диаграмма, схематически иллюстрирующая вариант, в котором линейные двухцепочечные фрагменты ДНК связывают друг с другом в соответствии с принципом осуществления способа получения ДНК согласно изобретению. Сначала, 3'→5'-экзонуклеаза 2 действует на линейный двухцепочечный ДНК-фрагмент 1a и на линейный фрагмент двухцепочечный ДНК 1b, которые имеют гомологичную область Н, в результате чего эта гомологичная область Н становится одноцепочечной. Белок рекомбиназа семейства RecA 3 действует на гомологичную область Н в одноцепочечной форме, и гомологичные области Н, комплементарные друг другу, подвергают гибридизации, в результате чего линейный двухцепочечный ДНК-фрагмент 1a и линейный фрагмент двухцепочечный ДНК 1b становятся одноцепочечными. Как показано справа на фиг. 1, расщепление под действием 3'→5'-экзонуклеазы 2 может быть осуществлено только на одном линейном двухцепочечном фрагменте ДНК 1а или на одном линейном двухцепочечном фрагменте ДНК 1b. Так, например, гомологичная область Н линейного двухцепочечного фрагмента ДНК 1a в одноцепочечной форме действует на гомологичную область Н линейного двухцепочечного фрагмента ДНК 1b в двухцепочечной форме в присутствии белка рекомбиназы семейства RecA 3, и оба эти фрагмента являются связанными.
[0019] При связывании линейных двухцепочечных фрагментов ДНК или при связывании линейного двухцепочечного фрагмента ДНК и одноцепочечного фрагмента ДНК, в способе получения ДНК согласно изобретению, двухцепочечный фрагмент ДНК расщепляют экзонуклеазой с образованием гомологичной области в одноцепочечной форме, а затем проводят реакцию связывания в присутствии белка рекомбиназы семейства RecA. По этой причине, способ получения ДНК согласно изобретению является превосходным с точки зрения эффективности связывания и позволяет осуществлять связывание множества линейных двухцепочечных фрагментов ДНК за одну реакцию, что трудно осуществить стандартными методами.
[0020] При связывании линейных одноцепочечных фрагментов ДНК в способе получения ДНК согласно изобретению, белок рекомбиназа семейства RecA быстро образует нити на каждом одноцепочечном фрагменте ДНК, что приводит к ингибированию расщепления экзонуклеазой. После этого, гомологичные области H, комплементарные друг другу, гибридизуются друг с другом под действием белка рекомбиназы семейства RecA, и оба одноцепочечных фрагмента ДНК являются связанными.
[0021] В способе получения ДНК согласно изобретению, число связанных фрагментов ДНК, предпочтительно равно 5 (5 фрагментов) или более, более предпочтительно 7 (7 фрагментов) или более, еще более предпочтительно 10 (10 фрагментов) или более, и может быть равно 20 (20 фрагментов) или более. Верхний предел числа связанных фрагментов ДНК в способе получения ДНК согласно изобретению не имеет конкретных ограничений. Так, например, связанными могут быть до 100 фрагментов. В способе получения ДНК согласно изобретению, например, приблизительно 50 линейных двухцепочечных фрагментов ДНК могут быть связаны путем оптимизации условий проведения реакции и т.п. В способе получения ДНК согласно изобретению, фрагменты ДНК могут быть связаны друг с другом и могут происходить от различных видов. Этот способ также позволяет связывать фрагменты ДНК, содержащие два или более фрагментов ДНК одного и того же типа.
[0022] Каждый связанный фрагмент ДНК двух или более типов согласно изобретению включает гомологичную область или комплементарную область для связывания по меньшей мере с одним из других фрагментов ДНК. В способе получения ДНК согласно изобретению, при связывании линейных двухцепочечных фрагментов ДНК или при связывании линейного двухцепочечного фрагмента ДНК и одноцепочечного фрагмента ДНК, сначала одну цепь двухцепочечного фрагмента ДНК расщепляют экзонуклеазой с получением гомологичной области в одноцепочечной форме. По этой причине, гомологичная область предпочтительно присутствует на конце линейного двухцепочечного фрагмента ДНК. Гомологичная область может присутствовать рядом с этим концом. Так, например, основание на концевой стороне линейного двухцепочечного фрагмента ДНК в конце гомологичной области, предпочтительно, находится в пределах 300 оснований от конца, более предпочтительно, в пределах 100 оснований, еще более предпочтительно, в пределах 30 оснований, а еще более предпочтительно, в пределах 10 оснований. С другой стороны, при связывании линейных одноцепочечных фрагментов ДНК, расщепление экзонуклеазой ингибируется под действием нитей белков рекомбиназы семейства RecA. Таким образом, комплементарная область может присутствовать в любой части одноцепочечного фрагмента ДНК.
[0023] Последовательности оснований гомологичных областей или комплементарных областей могут представлять собой одну и ту же последовательность оснований во всех связанных фрагментах ДНК. Предпочтительно, чтобы последовательности оснований гомологичных областей в связанных фрагментах ДНК отличались во фрагментах ДНК каждого типа по порядку из расположения. Так, например, для связывания двухцепочечного фрагмента ДНК А, двухцепочечного фрагмента ДНК В и двухцепочечного фрагмента ДНК С в указанном порядке, гомологичную область a помещают в нижерасположенный конец двухцепочечного фрагмента ДНК А и в вышерасположенный конец двухцепочечного фрагмента ДНК B, а гомологичную область b помещают в нижерасположенный конец двухцепочечного фрагмента ДНК В и в вышерасположенный конец двухцепочечного фрагмента ДНК С. Двухцепочечной фрагмент ДНК А и двухцепочечный фрагмент ДНК В связаны друг с другом в гомологичной области а. Двухцепочечной фрагмент ДНК В и двухцепочечный фрагмент ДНК С связаны друг с другом в гомологичной области b. Это позволяет получить линейную ДНК, в которой двухцепочечный фрагмент ДНК А, двухцепочечный фрагмент ДНК В и двухцепочечный фрагмент ДНК С связаны друг с другом в указанном порядке. В этом случае, гомологичная область с также находится в нижерасположенном конце двухцепочечного фрагмента ДНК С и в вышерасположенном конце двухцепочечного фрагмента ДНК А. В результате, двухцепочечный фрагмент ДНК А и двухцепочечный фрагмент ДНК В связываются в гомологической области а, двухцепочечный фрагмент ДНК В и двухцепочечный фрагмент ДНК С связываются в гомологической области b, а двухцепочечный фрагмент ДНК С и двухцепочечный фрагмент ДНК А связываются в гомологической области с. Это позволяет получить кольцевую ДНК, в которой двухцепочечный фрагмент ДНК А, двухцепочечный фрагмент ДНК В и двухцепочечный фрагмент ДНК С связаны в указанном порядке.
[0024] Гомологичная область и комплементарная область могут представлять собой любую последовательность, при условии, что отдельные цепи, имеющие такую область, могут специфически гибридизоваться друг с другом в реакционном растворе реакции связывания. Длина пар оснований (п.о.), отношение GC и т.п. в данной области могут быть соответствующим образом определены с применением общего метода конструирования зондов и праймеров. Вообще говоря, длина пар оснований гомологичной области должна быть достаточной для подавления неспецифической гибридизации и для правильного связывания представляющих интерес линейных двухцепочечных фрагментов ДНК. С другой стороны, если пары оснований гомологичной области являются слишком длинными, то это может снижать эффективность связывания. В настоящем изобретении, длина пары оснований гомологичной области или комплементарной области, предпочтительно, составляет 10 пар оснований (п.о.) или более, более предпочтительно 15 п.о. или более, а еще более предпочтительно 20 п.о. или более. Длина пары оснований гомологичной области или комплементарной области, предпочтительно, составляет 500 п.о. или менее, более предпочтительно 300 п.о. или менее, а еще более предпочтительно 200 п.о. или менее.
[0025] В способе получения ДНК согласно изобретению, длина фрагментов ДНК, связанных друг с другом, не имеет конкретных ограничений. Так, например, длина линейного двухцепочечного фрагмента ДНК, предпочтительно, составляет 50 п.о. или более, более предпочтительно, 100 п.о. или более, а еще более предпочтительно, 200 п.о. или более. Длина одноцепочечного фрагмента ДНК, предпочтительно, составляет 50 оснований или более, более предпочтительно, 100 оснований или более, а еще более предпочтительно, 200 оснований или более. В способе получения ДНК согласно изобретению могут быть также связаны двухцепочечные фрагменты ДНК размером 325 т.п.о. Длина связанных фрагментов ДНК может варьироваться в зависимости от их типов.
[0026] В способе получения ДНК согласно изобретению, вся гомологичная область или часть гомологичной области линейных двухцепочечных фрагментов ДНК, связанных друг с другом, должны иметь двухцепочечную структуру, в которой две одноцепочечные ДНК гибридизуются друг с другом. То есть, линейный двухцепочечный фрагмент ДНК может представлять собой полноразмерный линейный двухцепочечный фрагмент ДНК без гэпов или ников, либо он может представлять собой линейный двухцепочечный фрагмент ДНК, имеющий одноцепочечную структуру в одном или более положениях. Так, например, связанный линейный двухцепочечный фрагмент ДНК может иметь тупой конец или выступающий конец. В способе получения ДНК согласно изобретению могут быть связаны линейный двухцепочечный фрагмент ДНК с тупым концом и линейный двухцепочечный фрагмент ДНК с выступающим концом.
[0027] Молярное отношение каждого фрагмента ДНК, включенного в реакционный раствор, предпочтительно, является таким же, как и отношение числа молекул каждого фрагмента ДНК, составляющего представляющую интерес связанную молекулу. Путем спаривания определенного числа фрагментов ДНК в реакционной системе в начале реакции связывания, такая реакция связывания может быть осуществлена более эффективно. Так, например, при связывании фрагментов ДНК всех различных типов, молярные концентрации каждого фрагмента ДНК, содержащегося в реакционном растворе, предпочтительно являются одинаковыми.
[0028] Общее количество фрагментов ДНК, включенных в реакционный раствор, не имеет конкретных ограничений. Поскольку достаточное количество связанного продукта может быть легко получено, то общая концентрация фрагментов ДНК, содержащихся в реакционном растворе в начале реакции связывания, предпочтительно, составляет 0,01 нМ или более, более предпочтительно, 0,1 нМ или более, а еще более предпочтительно, 0,3 нM или более. Поскольку эффективность связывания является более высокой и подходящей для связывания множества фрагментов, то общая концентрация фрагментов ДНК, содержащихся в реакционном растворе в начале реакции связывания, предпочтительно, составляет 100 нМ или менее, более предпочтительно, 50 нМ или менее, еще более предпочтительно, 25 нМ или менее, а особенно предпочтительно, 20 нМ или менее.
[0029] В способе получения ДНК согласно изобретению, размер связанной молекулы, полученной посредством реакции связывания, не имеет конкретных ограничений. Так, например, размер полученной связанной молекулы, предпочтительно, составляет 1000 оснований или более, более предпочтительно, 5000 оснований или более, еще более предпочтительно, 10000 оснований или более, а еще более предпочтительно, 20000 оснований или более. Способ получения ДНК согласно изобретению позволяет получить связанную молекулу, имеющую длину 300000 оснований или более, предпочтительно, 500000 оснований или более, а еще более предпочтительно, 2000000 оснований или более.
[0030] Экзонуклеаза, используемая в настоящем изобретении, представляет собой фермент, который последовательно гидролизует линейную ДНК от 3'-конца или 5'-конца. Экзонуклеаза, используемая в настоящем изобретении, не имеет конкретных ограничений по типу или биологическому происхождению, при условии, что она будет обладать ферментативной активностью, обеспечивающей последовательный гидролиз от 3'-конца или 5'-конца линейной ДНК. Примерами ферментов, которые осуществляют последовательный гидролиз от 3'-конца (3'→5'-экзонуклеаза), является 3'→5'-экзонуклеаза, специфичная к линейной двухцепочечной ДНК, такая как экзонуклеаза семейства III-AP (непуриновая/непиримидиновая) эндонуклеаза и 3'→5'-экзонуклеаза, специфичная к одноцепочечной ДНК, такая как белок суперсемейства DnaQ. Примерами эндонуклеаз семейства III-AP-эндонуклеаз являются экзонуклеаза III (происходящая от Escherichia coli), ExoA (гомолог экзонуклеазы III Bacillus subtilis), Mth212 (гомолог экзонуклеазы III архебактерий), и АР-эндонуклеаза I (человеческий гомолог экзонуклеазы III). Примерами белков суперсемейства DnaQ являются экзонуклеаза I (происходящая от E. coli), экзонуклеаза Т (Exo Т) (также известная как РНКаза Т), экзонуклеаза X, субъединица эпсилон ДНК-полимеразы III, ДНК-полимераза I, ДНК-полимераза II, ДНК-полимераза Т7, ДНК-полимераза Т4, ДНК-полимераза Кленова 5, ДНК-полимераза Phi29, рибонуклеаза III (РНКаза D) и олигорибонуклеаза (ОRN). Примерами ферментов, которые осуществляют последовательный гидролиз линейной ДНК от 5'-конца (5'→3'-экзонуклеаза), являются λ-экзонуклеаза, экзонуклеаза VIII, экзонуклеаза Т5, экзонуклеаза Т7 и экзонуклеаза RecJ.
[0031] Предпочтительной экзонуклеазой, используемой в настоящем изобретении, является 3'→5'-экзонуклеаза с точки зрения эффективного баланса между способностью расщеплять линейные двухцепочечные фрагменты ДНК и эффективностью связывания в присутствии белков рекомбиназы семейства RecA. Среди этих экзонуклеаз, более предпочтительной является 3'→5'-экзонуклеаза, специфичная к линейной двухцепочечной ДНК, еще более предпочтительной является экзонуклеаза семейства III-АР-экзонуклеаз, а особенно предпочтительной является экзонуклеаза III.
[0032] В настоящем изобретении, реакционный раствор, предпочтительно, содержит 3'→5'-экзонуклеазу, специфичную к линейной двухцепочечной ДНК, и 3'→5'-экзонуклеазу, специфичную к одноцепочечной ДНК, в качестве экзонуклеаз. Эффективность связывания значительно улучшается в результате объединения 3'→5'-экзонуклеазы, специфичной к одноцепочечной ДНК, и 3'→5'-экзонуклеазы, специфичной к линейной двухцепочечной ДНК, по сравнению с эффективностью при использовании только 3'→5'-экзонуклеазы, специфичной к линейной двухцепочечной ДНК. Предполагается, что 3'-выступающий конец, образующийся в связанной молекуле под действием 3'→5'-экзонуклеазы, специфичной к линейной двухцепочечной ДНК и RecA, гидролизуется под действием 3'→5'-экзонуклеазы, специфичной к одноцепочечной ДНК, что приводит к повышению эффективности связывания. Поскольку эффективность связывания может быть значительно улучшена, то экзонуклеаза, содержащаяся в реакционном растворе согласно изобретению, предпочтительно, представляет собой комбинацию экзонуклеазы семейства III-АР-эндонуклеазы и 3'→5'-экзонуклеазы, специфичной к одноцепочечной ДНК одного или более типов. Более предпочтительной является комбинация экзонуклеазы семейства III-АР-эндонуклеазы и белков суперсемейства DnaQ одного или более типов. Особенно предпочтительной является комбинация экзонуклеазы III и экзонуклеазы I или комбинация экзонуклеазы III, экзонуклеазы I и экзонуклеазы Т.
[0033] В настоящем изобретении, концентрация экзонуклеазы в реакционном растворе для проведения реакции связывания в начале реакции связывания, предпочтительно, составляет, например, 1-1000 мЕд/мкл, более предпочтительно, 5-1000 мЕд/мкл, более предпочтительно, 5-500 мЕд/мкл, а еще более предпочтительно, 10-150 мЕд/мкл. В частности, если экзонуклеазой является 3'→5'-экзонуклеаза, специфичная к линейной двухцепочечной ДНК, то концентрация 3'→5'-экзонуклеазы, специфичной к линейной двухцепочечной ДНК, в реакционном растворе в начале реакции связывания составляет, предпочтительно, например, 5-500 мЕд/мкл, более предпочтительно, 5-250 мЕд/мкл, более предпочтительно, 5-150 мЕд/мкл, а еще более предпочтительно, 10-150 мЕд/мкл. Если экзонуклеазой является 3'→5'-экзонуклеаза, специфичная к одноцепочечной ДНК, то концентрация 3'→5'-экзонуклеазы, специфичной к одноцепочечной ДНК, в реакционном растворе в начале реакции связывания составляет, предпочтительно, например, 1-10000 мЕд/мкл, более предпочтительно, 100-5000 мЕд/мкл, а еще более предпочтительно, 200-2000 мЕд/мкл. Если 3'→5'-экзонуклеаза, специфичная к линейной двухцепочечной ДНК, и 3'→5'-экзонуклеаза, специфичная к одноцепочечной ДНК, используются в комбинации, то концентрация каждой экзонуклеазы в реакционном растворе, в начале реакции связывания может представлять собой предпочтительную концентрацию каждой из вышеуказанных экзонуклеаз.
[0034] В настоящем изобретении и в настоящем описании, белок рекомбиназа семейства RecA означает белок, обладающий рекомбиназной активностью семейства RecA. Эта активность включает функцию полимеризации на одноцепочечной или двухцепочечной ДНК с образованием нити; гидролитическую активность для нуклеозид-трифосфатов, таких как АТФ (аденозин-трифосфат) и функцию поиска гомологичной области и осуществления гомологичной рекомбинации. Примерами белков рекомбиназы семейства RecA являются гомолог RecA прокариотов, гомолог RecA бактериофага, гомолог RecA архебактерий, гомолог RecA эукариотов и т.п. Примерами гомологов RecA прокариотов являются RecA E. coli; RecA, происходящий от в высокой степени термофильных бактерий, таких как бактерии Thermus, такие как Thermus thermophiles и Thermus aquaticus, бактерии Thermococcus, бактерии Pyrococcus, и бактерии Thermotoga; RecA, происходящий от бактерий, резистентных к облучению, таких как Deinococcus radiodurans. Примерами гомологов RecA бактериофага является фаг T4 UvsX. Примерами гомологов RecA архебактерий является RadA. Примерами гомологов RecA эукариотов являются Rad51 и его паралог, и Dcm1. Аминокислотные последовательности этих гомологов RecA могут быть взяты из баз данных, таких как NCBI (http://www.ncbi.nlm.nih.gov/).
[0035] Белок рекомбиназа семейства RecA, используемый в настоящем изобретении, может представлять собой белок дикого типа или его вариант. Вариант представляет собой белок дикого типа, в который были введены одна или более мутаций, а именно, делеции, добавления или замены 1-30 аминокислот, и который сохраняет рекомбиназную активность семейства RecA. Примерами вариантов являются варианты с аминокислотными заменами, которые улучшают функцию поиска гомологичных областей в белках дикого типа; варианты с различными метками, добавленными к N-концу или С-концу белков дикого типа, и варианты с повышенной терморезистентностью (WO 2016/013592). В качестве метки, например, могут быть использованы метки, широко применяемые для экспрессии или очистки рекомбинантных белков, такие как His-метка, HA-метка (гемагглютининовая метка), метка Myc и метка Flag. Термин «белок рекомбиназы семейства RecA дикого типа» означает белок, имеющий такую же аминокислотную последовательность, как и аминокислотная последовательность белка рекомбиназы семейства RecA, который сохранялся в микроорганизмах, выделенных из природных источников.
[0036] Белок рекомбиназа семейства RecA, используемый в настоящем изобретении, предпочтительно, представляет собой вариант, который сохраняет белок рекомбиназу семейства RecA. Примерами вариантов являются мутант F203W, в котором 203-й аминокислотный остаток фенилаланин RecA E.coli заменен триптофаном, и мутанты, в которых фенилаланин, соответствующий 203 фенилаланину RecA E. coli, заменен триптофаном в различных гомологах RecA.
[0037] В настоящем изобретении, количество белка рекомбиназы семейства RecA в реакционном растворе для реакции связывания не имеет конкретных ограничений. В настоящем изобретении, концентрация белка рекомбиназы семейства RecA в реакционном растворе в начале реакции связывания предпочтительно, составляет, например, 0,01-100 мкМ, более предпочтительно, 0,1-100 мкМ, еще более предпочтительно, 0,1-50 мкМ, еще более предпочтительно, 0,5-10 мкМ, а особенно предпочтительно, 1,0-5,0 мкМ.
[0038] Нуклеозид-трифосфаты или дезоксинуклеозид-трифосфаты неоходимы для сообщения рекомбиназной активности семейства RecA белкам рекомбиназы семейства RecA. По этой причине, реакционный раствор для реакции связывания согласно изобретению содержит по меньшей мере один нуклеозид-трифосфат или дезоксинуклеотид-трифосфат. Поскольку нуклеозид-трифосфат содержится в реакционном растворе для реакции связывания согласно изобретению, то предпочтительно, использовать одно или более соединений, выбранных из группы, состоящей из dATP (дезоксиаденозин-трифосфата), dGTP (дезоксигуанозин-трифосфата), dСTP (дезоксицитидин-трифосфата) и dTTP (дезокситимидин-трифосфата). Особенно предпочтительно использовать dATP. Общее количество нуклеозид-трифосфата и дезоксинуклеотид-трифосфата, содержащихся в реакционном растворе, не имеет конкретных ограничений, при условии, что оно будет достаточным для того, чтобы белок рекомбиназа семейства RecA обладал рекомбиназной активностью семейства RecA. В настоящем изобретении, концентрация нуклеозид-трифосфата или концентрация дезоксинуклеотид-трифосфата в реакционном растворе для проведения реакции связывания в начале этой реакции связывания, предпочтительно, составляет, например, 1 мкМ или более, более предпочтительно, 10 мкМ или более, а еще более предпочтительно, 30 мкМ или более. С другой стороны, если концентрация нуклеозид-трифосфата в реакционном растворе является слишком высокой, то эффективность связывания множества фрагментов может быть немного снижена. Поэтому, концентрация нуклеозид-трифосфата или концентрация дезоксинуклеотид-трифосфата в реакционном растворе в начале реакции связывания, предпочтительно, составляет, например, 1000 мкМ или менее, более предпочтительно, 500 мкМ или менее, а еще более предпочтительно, 300 мкМ или менее.
[0039] Ионы магния (Mg2+) необходимы для сообщения белку рекомбиназе семейства RecA рекомбиназной активности семейства RecA и для сообщения экзонуклеазе экзонуклеазной активности. Следовательно, реакционный раствор для реакции связывания согласно изобретению содержит источник ионов магния. Источник ионов магния представляет собой вещество, которое передает ионы магния в реакционный раствор. Примерами являются соли магния, такие как ацетат магния [Mg(OAc)2], хлорид магния [MgCl2] и сульфат магния [MgSO4]. Предпочтительным источником ионов магния является ацетат магния.
[0040] В настоящем изобретении, концентрация источника ионов магния в реакционном растворе для реакции связывания не имеет конкретных ограничений, при условии, что белок рекомбиназа семейства RecA будет обладать рекомбиназной активностью семейства RecA, а экзонуклеаза будет обладать экзонуклеазной активностью. Концентрация источника ионов магния в реакционном растворе в начале реакции связывания предпочтительно, составляет, например, 0,5 мМ или более, а более предпочтительно 1 мМ или более. С другой стороны, если концентрация ионов магния в реакционном растворе является слишком высокой, то экзонуклеазная активность также становится слишком высокой, и это может снижать эффективность связывания множества фрагментов. По этой причине, концентрация источника ионов магния в реакционном растворе в начале реакции связывания, предпочтительно, составляет, например, 20 мМ или менее, более предпочтительно, 15 мМ или менее, еще более предпочтительно, 12 мМ или менее, а наиболее предпочтительно, 10 мМ или менее.
[0041] Реакционный раствор для реакции связывания согласно изобретению получают, например, путем добавления фрагментов ДНК, белка рекомбиназы семейства RecA, экзонуклеазы, по меньшей мере одного нуклеозид-трифосфата или дезоксинуклеотид-трифосфата и источника ионов магния в буфер. Буферный раствор не имеет конкретных ограничений, при условии, что он будет подходящим для использования при рН 7-9, а предпочтительно, рН 8. Примерами буферов являются Трис-HCl, Трис-ОАс, Hepes-КОН, фосфатный буфер, MOPS-NaOH, и трицин-HCl. Предпочтительным буфером является Трис-HCl или Трис-ОАс. Концентрация буферного раствора не имеет конкретных ограничений и может быть соответствующим образом выбрана специалистом в данной области. В случае Трис-HCl или Трис-OAc, например, концентрация буферного раствора может быть выбрана из концентрации 10 мМ - 100 мМ, предпочтительно, 10 мМ -50 мМ, а более предпочтительно, 20 мМ.
[0042] В настоящем изобретении, если в качестве белка рекомбиназы семейства RecA используется UvsX, то предпочтительно, чтобы реакционный раствор для реакции связывания также содержал UvsY фага Т4. UvsY является медиатором гомологической рекомбинации в фаге Т4. В фаге Т4, сначала одноцепочечная ДНК связывается с gp32 (с белком, связывающимся с одноцепочечной ДНК) с образованием комплекса «одноцепочечная ДНК-gp32». Затем, одноцепочечная ДНК связывается с uvsX, в результате чего, gp32 в комплексе заменяется на uvsX, что приводит к гомологической рекомбинации. UvsY стимулирует связывание одноцепочечной ДНК и uvsX посредством дестабилизации взаимодействия одноцепочечной ДНК-gp32 и стабилизации взаимодействия одноцепочечной ДНК-uvsX, что приводит к ускорению реакции гомологичной рекомбинации (Bleuit et al., Proceedings of the National Academy of Sciences of the United States of America, 2001, vol.98 (15), p.8298-8305). Кроме того, в настоящем изобретении, эффективность связывания также стимулируется с использованием UvsY вместе с UvsX.
[0043] Реакционный раствор для реакции связывания согласно изобретению содержит фрагмент ДНК, белок рекомбинантного фермента семейства RecA, экзонуклеазу, по меньшей мере нуклеозид-трифосфат или дезоксинуклеотид-трифосфат, и источник ионов магния. Реакционный раствор также предпочтительно содержит регенерирующий фермент для нуклеозид-трифосфата или дезоксинуклеотид-трифосфата и его субстрат. Благодаря регенерации нуклеозид-трифосфатов или дезоксинуклеотид-трифосфатов в реакционном растворе, большое количество фрагментов ДНК может связываться более эффективно. Примерами комбинаций регенерирующих ферментов и их субстратов для регенерации нуклеозид-трифосфата или дезоксинуклеотид-трифосфата являются комбинация креатинкиназы и фосфата креатина, комбинация пируват-киназы и пирувата фосфоэнола, комбинация ацетат-киназы и ацетилфосфата, комбинация полифосфат-киназы и полифосфата, и комбинация нуклеозид-дифосфаткиназы и нуклеозид-трифосфата. Нуклеозид-трифосфат, используемый в качестве субстрата (источник пополнения фосфата) для нуклеозид-дифосфаткиназы может представлять собой любой из ATP, GTP, CTP и UTP. Кроме того, примером регенерирующего фермента является миокиназа.
[0044] В настоящем изобретении, концентрации регенерирующего фермента для нуклеозид-трифосфата и его субстрата в реакционном растворе для реакции связывания не имеют конкретных ограничений, при условии, что эти концентрации будут достаточными для регенерации нуклеозид-трифосфата во время проведения реакции связывания в реакционном растворе. Так, например, при использовании креатинкиназы и фосфата креатина, концентрация креатинкиназы в реакционном растворе для реакции связывания согласно изобретению, предпочтительно, составляет 1-1000 нг/мкл, более предпочтительно 5-1000 нг/мкл, еще более предпочтительно 5-500 нг/мкл, а еще более предпочтительно 5-250 нг/мкл. Концентрация фосфата креатина в растворе, предпочтительно, составляет 0,4-20 мМ, более предпочтительно 0,4-10 мМ, а еще более предпочтительно 1-7 мМ.
[0045] Если множество фрагментов связаны в нужном порядке, то последовательности оснований гомологичной области или комплементарной области, предпочтительно, отличаются для каждой комбинации связанных фрагментов ДНК. Однако, при тех же температурных условиях, одноцепочечная гомологичная область, имеющая высокое содержание G (гуанинового основания) и С (цитозинового основания), имеет тенденцию к образованию вторичной структуры. С другой стороны, гомологичная область, имеющая высокое содержание А (аденинового основания) и Т (тиминового основания), обладает низкой эффективностью гибридизации. Таким образом, эффективность связывания может снижаться. При ингибировании образования вторичной структуры одноцепочечной ДНК и стимуляции специфической гибридизации может быть повышена стимуляция связывания фрагментов ДНК.
[0046] Поэтому, предпочтительно, чтобы было добавлено вещество, которое ингибирует образование вторичной структуры одноцепочечной ДНК и стимулирует специфическую гибридизацию в реакционном растворе для реакции связывания согласно изобретению. Примерами таких веществ являются диметилсульфоксид (ДМСО) и хлорид тетраметиламмония (ТМАС). ДМСО подавляет образование вторичной структуры пар оснований, богатых GC. ТМАС стимулирует специфическую гибридизацию. В настоящем изобретении, при добавлении в реакционный раствор для реакции связывания вещества, которое стимулирует образование вторичной структуры одноцепочечной ДНК и стимулирует специфическую гибридизацию, концентрация такого вещества не имеет конкретных ограничений, при условии, что с использованием такого вещества будет достигаться эффект стимуляции связывания фрагментов ДНК. Так, например, при использовании ДМСО в качестве такого вещества, концентрация ДМСО в реакционном растворе для реакции связывания согласно изобретению составляет, предпочтительно, 5-30% по объему, более предпочтительно, 8-25% по объему, а еще более предпочтительно, 8-20% по объему. При использовании ТМАС в качестве такого вещества, концентрация ТМАС в реакционном растворе для реакции связывания согласно изобретению составляет, предпочтительно, 60-300 мМ, более предпочтительно, 100-250 мМ, а еще более предпочтительно, 100-200 мМ.
[0047] В настоящем изобретении, к реакционному раствору для реакции связывания, предпочтительно, чтобы было добавлено вещество, сообщающее эффект макромолекулярной полимеризации. Эффект макромолекулярной полимеризации может стимулировать взаимодействие молекул ДНК друг с другом и связывание фрагментов ДНК. Примерами таких веществ являются полиэтиленгликоль (ПЭГ) 200-20000, поливиниловый спирт (ПВС) 200-20000, декстран 40-70, Фиколл 70 и альбумин бычьей сыворотки (BSA). В настоящем изобретении, при добавлении вещества, сообщающего эффект макромолекулярной полимеризации, в реакционный раствор для реакции связывания, концентрация такого вещества не имеет конкретных ограничений, при условии, что это вещество будет стимулировать связывание фрагментов ДНК. Так, например, при использовании ПЭГ8000 в качестве такого вещества, концентрация этого вещества в реакционном растворе для реакции связывания составляет, предпочтительно, 2-20% по массе, более предпочтительно, 2-10% по массе, а еще более предпочтительно, 4-6% по массе.
[0048] В настоящем изобретении, реакционный раствор для реакции связывания может также содержать источник ионов щелочного металла. Источник ионов щелочного металла представляет собой вещество, которое вводит в реакционный раствор ионы щелочного металла. В настоящем изобретении, ион щелочного металла, содержащийся в реакционном растворе для реакции связывания, предпочтительно, представляет собой ион натрия (Na+) или ион калия (K+). Примерами источника ионов щелочного металла являются глутамат калия [KGlu], аспартат калия, хлорид калия, ацетат калия [КОАс], глутамат натрия, аспартат натрия, хлорид натрия и ацетат натрия. В настоящем изобретении, источник ионов щелочного металла, содержащийся в реакционном растворе для реакции связывания, предпочтительно, представляет собой глутамат калия или ацетат калия. Глутамат калия является особенно предпочтительным, поскольку он повышает эффективность связывания множества фрагментов. Концентрация источника ионов щелочного металла в реакционном растворе в начале реакции связывания не имеет конкретных ограничений. Так, например, концентрация источника ионов щелочного металла, предпочтительно, представляет собой концентрацию, которая может быть скорректирована до значения концентрации ионов щелочного металла в реакционном растворе, составляющего 10 мМ или более, предпочтительно, в пределах 30-300 мМ, а еще более предпочтительно, в пределах 50-150 мМ.
[0049] В настоящем изобретении, реакционный раствор для реакции связывания может также содержать восстановитель. Примерами восстановителя являются дитиотреитол (DТТ), β-меркаптоэтанол (2-меркаптоэтанол), трис(2-карбоксиэтил)фосфин (ТСЕР) и глутатион. Предпочтительным восстановителем является DТТ. Восстановитель может содержаться в реакционном растворе в концентрации 1,0-15,0 мМ, а предпочтительно, 2,0-10,0 мМ.
[0050] В способе получения ДНК согласно изобретению, реакцию связывания осуществляют путем инкубирования реакционного раствора в течение предварительно определенного периода времени в изотермических условиях при температуре, при которой белок рекомбинантного фермента семейства RecA и экзонуклеаза в реакционном растворе могут обладать ферментативной активностью. Реакционный раствор получают в буферном растворе, содержащем фрагменты ДНК двух или более видов, белок рекомбинантного фермента семейства RecA, нуклеозид-трифосфат и источник ионов магния, и кроме того, если это необходимо, экзонуклеазу, набор ферментов для регенерации нуклеозид-трифосфата и их субстратов; вещество, которое подавляет образование вторичной структуры одноцепочечной ДНК и стимулирует специфическую гибридизацию; вещество, сообщающее эффект макромолекулярной полимеризации; источник ионов щелочного металла и восстановитель. Температура реакции связывания, предпочтительно, составляет в пределах 25-48°С, а более предпочтительно, в пределах 27-45°С. В частности, если длина гомологичной области или комплементарной области составляет 50 оснований или более, то температура реакции связывания, предпочтительно, составляет в пределах 30-45°С, более предпочтительно, в пределах 37-45°С, а еще более предпочтительно, в пределах 40-43°С. С другой стороны, если длина гомологичной области или комплементарной области составляет 50 оснований или менее, то температура реакции связывания, предпочтительно, составляет в пределах 27-43°С, более предпочтительно, в пределах 27-37°С, а еще более предпочтительно, в пределах 27-33°С. В настоящем описании, термин «изотермические условия» означает условия, при которых температура во время реакции поддерживается в пределах от ±3°С или ±1°С. Время реакции связывания не имеет конкретных ограничений и может составлять, например, от 15 минут до 6 часов, а предпочтительно, от 15 минут до 2 часов.
[0051] Как показано на фиг. 1, в связанной молекуле (линейной или кольцевой ДНК), полученной посредством реакции связывания, присутствуют гэпы и ники. Гэп означает отсутствие одного или более смежных нуклеотидов в двухцепочечной ДНК. Ник означает разрыв, образующийся в результате расщепления фосфодиэфирной связи между соседними нуклеотидами в двухцепочечной ДНК. В способе получения ДНК согласно изобретению, после реакции связывания, предпочтительно, осуществлять репарацию гэпов и ников в полученной связанной молекуле с использованием ферментов для репарации гэпов и dNTP. После репарации гэпов и ников, связанная молекула может быть превращена в полноразмерную двухцепочечную ДНК.
[0052] В частности, репарацию и ников связанной молекулы осуществляют путем добавления ферментов для репарации гэпов и dNTP в реакционный раствор после реакции связывания с последующим инкубированием в течение предварительно определенного периода времени в изотермических условиях, при которых группа ферментов для репарации гэпов будет обладать ферментативной активностью. Типы ферментов для репарации гэпов не имеют конкретных ограничений и могут иметь любое биологическое происхождение, при условии, что они будут обладать способностью репарировать гэпы и ники в двухцепочечной ДНК. В качестве ферментов для репарации гэпов, например, может быть использована комбинация фермента, обладающего ДНК-полимеразной активностью, и фермента, обладающего ДНК-лигазной активностью. При использовании ДНК-лигазы, происходящей от Escherichia coli, в качестве ДНК-лигазы, в реакционном растворе содержится NAD (никотинамид-аденин-динуклеотид), который является ее кофактором и содержится в концентрации в пределах 0,01-1,0 мМ. Обработка ферментами для репарации гэпов может быть осуществлена, например, при 25-40° в течение 5-120 минут, а предпочтительно, в течение 10-60 минут.
[0053] dNTP является общим термином для dATP, dGTP, dСTP и dTTP. Концентрация dNTP, содержащегося в реакционном растворе в начале реакции репарации, может составлять, например, в пределах 0,01-1 мМ, а предпочтительно, в пределах 0,05-1 мМ.
[0054] Также предпочтительно, чтобы связанная молекула (линейная или кольцевая ДНК) была дополнительно амплифицирована с репарацией гэпов и ников. Способ амплификации связанной молекулы с репарацией гэпов и ников не имеет конкретных ограничений. Амплификация может быть осуществлена общим методом амплификации с использованием линейной или кольцевой ДНК в качестве матрицы.
[0055] В способе получения ДНК согласно изобретению, если связанная молекула, полученная путем осуществления реакции репарации гэпов и ников после реакции связывания, является линейной, то связанную молекулу, предпочтительно, амплифицируют посредством полимеразной цепной реакции (ПЦР). ПЦР может быть осуществлена стандартным методом.
[0056] В способе получения ДНК согласно изобретению, если связанная молекула, полученная путем осуществления реакции репарации гэпов и ников после реакции связывания, является кольцевой, то связанную молекулу, предпочтительно, амплифицируют методом амплификации по типу «катящегося кольца» (RCA). RCA может быть осуществлена стандартным методом.
[0057] В способе получения ДНК согласно изобретению, если связанная молекула, полученная путем осуществления реакции репарации гэпов и ников после реакции связывания, является кольцевой и имеет последовательность ориджина репликации (ориджина хромосомы (Oric)), способную связываться с ферментом, обладающим активностью DnaA, то связанную молекулу, предпочтительно, амплифицируют методом амплификации посредством реакции репликативного цикла (RCR). При проведении RCR-амплификации с использованием связанной молекулы, полученной непосредственно после реакции связывания в качестве матрицы, то есть, без проведения реакции репарации гэпов и ников, можно получить кольцевую связанную молекулу, состоящую из полноразмерной двухцепочечной ДНК без гэпов и ников в качестве продукта амплификации.
[0058] Так, например, известная последовательность ориджина репликации, присутствующая в бактериях, таких как Escherichia coli и Bacillus subtilis, может быть взята из общедоступной базы данных, такой как NCBI. Последовательность ориджина репликации может быть также получена путем клонирования фрагмента ДНК, который может связываться с ферментом, обладающим активностью DnaA, и анализа его последовательности оснований.
[0059] В частности, метод RCR-амплификации может быть проведен путем получения реакционной смеси и инкубирования полученной реакционной смеси. Реакционная смесь включает кольцевую связанную молекулу, полученную путем реакции связывания, в качестве матрицы; первую группу ферментов, которые катализируют репликацию кольцевой ДНК; вторую группу ферментов, которые катализируют реакцию связывания фрагментов Оказаки и синтезируют две сестринских кольцевых ДНК, состоящих из катенана; третью группу ферментов, которые катализируют реакцию разделения двух сестринских кольцевых ДНК, и dNTP. Две сестринских кольцевых ДНК, состоящих из катенана, представляют собой две кольцевые ДНК, синтезированные посредством репликации ДНК и находящиеся в связанной форме.
[0060] Примером первой группы ферментов, которые катализируют репликацию кольцевой ДНК, является группа ферментов, представленная Kaguni J.M. и Kornberg А. Cell 1984, 38: 183-90. В частности, примерами первой группы ферментов являются один или более ферментов или групп ферментов, выбранных из группы, состоящей из фермента, обладающего активностью DnaA;, нуклеоидных белков одного или более типов; фермента или группы ферментов, обладающих ДНК-гиразной активностью; белка, связывающегося с одноцепочечной ДНК (SSB); фермента, обладающего геликазной активностью типа DnaB; фермента, обладающего ДНК-геликазной загрузочной активностью; фермента, обладающего ДНК-примазной активность; фермента, обладающего ДНК-захватывающей активностью; фермента или группы ферментов, обладающих активностью ДНК-полимеразы III*, и комбинаций всех вышеупомянутых ферментов или групп ферментов.
[0061] Биологическое происхождение фермента, обладающего активностью DnaA, не имеет конкретных ограничений, при условии, что такой фермент будет обладать инициирующей активностью, сходной с активностью DnaA, который представляет собой инициирующий белок Escherichia coli, при этом, предпочтительно, используется DnaA, происходящий от Escherichia coli. DnaA, происходящий от Escherichia coli, может присутствовать в качестве мономера в реакционном растворе в концентрации, составляющей, но не ограничивающейся ими, 1 нМ - 10 мМ, а предпочтительно, в концентрации 1 нМ - 5 мМ, 1 нМ - 3 мМ, 1 нМ - 1,5 мМ, 1 нМ - 1,0 мM, 1 нМ - 500 нМ, 50 нМ - 200 нМ или 50 нм - 150 нМ.
[0062] Нуклеотидный белок представляет собой белок в нуклеотиде. Биологическое происхождение нуклеотидного белка одного или более типов, используемого в настоящем изобретении, не имеет конкретных ограничений, при условии, что он будет обладать активностью, аналогичной активности нуклеотидного белка Escherichia coli. Так, например, предпочтительно используется IHF, происходящий от Escherichia coli, а именно, комплекс IhfA и/или IhfB (гетеродимер или гомодимер), или HU, происходящий от Escherichia coli, а именно, комплекс HupА и HupB. IHF, происходящий от Escherichia coli, может содержаться в качестве гетеро/гомодимера в реакционном растворе в концентрации в пределах 5 нМ-400 нМ. Предпочтительно, IHF, происходящий от Escherichia coli, может содержаться в реакционном растворе в концентрации в пределах 5 нМ-200 нМ, 5 нМ-100 нМ, 5 нМ-50 нМ, 10 нМ-50 нМ, 10 нМ-40 нМ или 10 нМ-30 нМ, хотя эти концентрации не ограничиваются вышеуказанными пределами. HU, происходящий от Escherichia coli, может содержаться в реакционном растворе в концентрации в пределах 1 нМ - 50 нМ, а предпочтительно, он может содержаться в этом растворе в концентрации в пределах 5 нМ - 50 нМ, или 5 нМ - 25 нМ, хотя эти концентрации не ограничиваются вышеуказанными пределами.
[0063] Биологическое происхождение фермента или группы ферментов, обладающих ДНК-гиразной активностью, не имеет конкретных ограничений, при условии, что эти ферменты будут обладать активностью, сходной с активностью ДНК-гиразы Escherichia coli. Так, например, предпочтительно, используется комплекс GyrА и GyrB, происходящих от Escherichia coli. Такой комплекс GyrА и GyrB, происходящих от Escherichia coli, может содержаться в виде гетеротетрамера в реакционном растворе в концентрации в пределах 20 нМ - 500 нМ, а предпочтительно, он может содержаться в реакционном растворе в концентрации в пределах 20 нМ - 400 нМ, 20 нМ - 300 нМ, 20 нМ - 200 нМ, 50 нМ - 200 нМ или 100 нМ - 200 нМ, однако, эти концентрации не ограничиваются вышеуказанными пределами.
[0064] Биологическое происхождение белка, связывающегося с одноцепочечной молекулой (SSB), не имеет конкретных ограничений, при условии, что он будет обладать активностью, аналогичной активности белка, связывающегося с одноцепочечной молекулой Escherichia coli. Так, например, предпочтительно, используется SSB, происходящий от Escherichia coli. Такой SSB, происходящий от Escherichia coli, может присутствовать в виде гомотетрамера в реакционном растворе в концентрации в пределах 20 нМ - 1000 нМ, а предпочтительно, он может присутствовать в этом растворе в концентрации в пределах 20 нМ - 500 нМ, 20 нМ - 300 нМ, 20 нМ - 200 нМ, 50 нМ - 500 нМ, 50 нМ - 400 нМ, 50 нМ - 300 нМ, 50 нМ - 200 нМ, 50 нМ - 150 нМ, 100 нМ - 500 нМ, или 100 нМ - 400 нМ, однако, эти концентрации не ограничиваются вышеуказанными пределами.
[0065] Биологическое происхождение фермента, обладающего геликазной активностью типа DnaВ, не имеет конкретных ограничений, при условии, что такой фермент будет обладать активностью, аналогичной активности DnaВ Escherichia coli. Так, например, предпочтительно, используется DnaВ, происходящий от Escherichia coli. Такой DnaВ, происходящий от Escherichia coli, может присутствовать в виде гомогексамера в реакционном растворе в концентрации в пределах 5 нМ - 200 нМ, а предпочтительно, он может присутствовать в этом растворе в концентрации в пределах 5 нМ - 100 мМ, 5 нМ - 50 мМ или 5 нМ - 30 нМ, однако, эти концентрации не ограничиваются вышеуказанными пределами.
[0066] Биологическое происхождение фермента, обладающего ДНК-геликазной загрузочной активностью, не имеет конкретных ограничений, при условии, что такой фермент будет обладать активностью, аналогичной активности DnaС Escherichia coli. Так, например, предпочтительно, используется DnaС, происходящий от Escherichia coli. Такой DnaС, происходящий от Escherichia coli, может присутствовать в виде гомогексамера в реакционном растворе в концентрации в пределах 5 нМ - 200 нМ, а предпочтительно, он может присутствовать в этом растворе в концентрации в пределах 5 нМ - 100 нМ, 5 нМ - 50 нМ или 5 нМ - 30 нМ, однако, эти концентрации не ограничиваются вышеуказанными пределами.
[0067] Биологическое происхождение фермента, обладающего ДНК-примазной активностью, не имеет конкретных ограничений, при условии, что такой фермент будет обладать активностью, аналогичной активности DnaG Escherichia coli. Так, например, предпочтительно, используется DnaG, происходящий от Escherichia coli. Такой DnaG, происходящий от Escherichia coli, может присутствовать в виде мономера в реакционном растворе в концентрации в пределах 20 нМ - 1000 нМ, а предпочтительно, он может присутствовать в этом растворе в концентрации в пределах 20 нМ - 800 нМ, 50 нМ - 800 нМ, 100 нМ - 800 нМ, 200 нМ - 800 нМ, 250 нМ - 800 нМ, 250 нМ - 500 нМ или 300 нМ - 500 нМ, однако, эти концентрации не ограничиваются вышеуказанными пределами.
[0068] Биологическое происхождение фермента, обладающего ДНК-захватывающей активностью, не имеет конкретных ограничений, при условии, что такой фермент будет обладать активностью, аналогичной активности DnaN Escherichia coli. Так, например, предпочтительно, используется DnaN, происходящий от Escherichia coli. Такой DnaN, происходящий от Escherichia coli, может присутствовать в виде гомодимера в реакционном растворе в концентрации в пределах 10 нМ - 1000 нМ, а предпочтительно, он может присутствовать в этом растворе в концентрации в пределах 10 нМ - 800 нМ, 10 нМ - 500 нМ, 20 нМ - 500 нМ, 20 нМ - 200 нМ, 30 нМ - 200 нМ или 30 нМ - 100 нМ, однако, эти концентрации не ограничиваются вышеуказанными пределами.
[0069] Биологическое происхождение фермента или группы ферментов, обладающих активностью ДНК-полимеразы III*, не имеет конкретных ограничений, при условии, что такой фермент или группа ферментов будут обладать активностью, аналогичной активности комплекса ДНК-полимеразы III* Escherichia coli. Так, например, предпочтительно, используется группа ферментов, включающая любые ферменты DnaX, HolA, HolB, HoLC, HoLD, DnaE, DnaQ и HolE, происходящие от Escherichia coli, предпочтительно, группа ферментов, включающая комплекс ферментов DnaX, HolA, HolB и DnaE, происходящих от Escherichia coli, а более предпочтительно, группа ферментов, включающая комплекс ферментов DnaX, HolA, HolB, HoLC, HoLD, DnaE, DnaQ и HolE, происходящих от Escherichia coli. Такой комплекс ДНК-полимеразы III*, происходящий от Escherichia coli, может присутствовать в виде гетеромультимера в реакционном растворе в концентрации в пределах 2 нМ - 50 нМ, а предпочтительно, он может присутствовать в этом растворе в концентрации в пределах 2 нМ - 40 нМ, 2 нМ - 30 нМ, 2 нМ - 20 нМ, 5 нМ - 40 нМ, 5 нМ - 30 нМ или 5 нМ - 20 нМ, однако, эти концентрации не ограничиваются вышеуказанными пределами.
[0070] Примерами вторых групп ферментов, которые катализируют созревание фрагмента Оказаки и синтезируют две сестринских кольцевых ДНК, состоящих из катенана, могут быть, например, один или более ферментов, выбранных из группы, состоящей из фермента, обладающего активностью ДНК-полимеразы I; фермента, обладающего ДНК-лигазной активностью; и фермента, обладающего активностью РНКазы H, или комбинации этих ферментов.
[0071] Биологическое происхождение фермента, обладающего активностью ДНК-полимеразы I, не имеет конкретных ограничений, при условии, что такой фермент будет обладать активностью, аналогичной активности ДНК-полимеразы I Escherichia coli. Так, например, предпочтительно, используется ДНК-полимераза I, происходящая от Escherichia coli. Такая ДНК-полимераза I, происходящая от Escherichia coli, может присутствовать в виде мономера в реакционном растворе в концентрации в пределах 10 нМ - 200 нМ, а предпочтительно, она может присутствовать в этом растворе в концентрации в пределах 20 нМ - 200 нМ, 20 нМ - 150 нМ, 20 нМ - 100 нМ, 40 нМ - 150 нМ, 40 нМ - 100 нМ или 40 нМ - 80 нМ, однако, эти концентрации не ограничиваются вышеуказанными пределами.
[0072] Биологическое происхождение фермента, обладающего ДНК-лигазной активностью, не имеет конкретных ограничений, при условии, что такой фермент будет обладать активностью, аналогичной активности ДНК-лигазы Escherichia coli. Так, например, предпочтительно, используется ДНК-лигаза, происходящая от Escherichia coli или ДНК-лигаза фага Т4. Такая ДНК-лигаза, происходящая от Escherichia coli, может присутствовать в виде мономера в реакционном растворе в концентрации в пределах 10 нМ - 200 нМ, а предпочтительно, она может присутствовать в этом растворе в концентрации в пределах 15 нМ - 200 нМ, 20 нМ - 200 нМ, 20 нМ - 150 нМ, 20 нМ - 100 нМ или 20 нМ - 80 нМ, однако, эти концентрации не ограничиваются вышеуказанными пределами.
[0073] Биологическое происхождение фермента, обладающего активностью РНКазы H, не имеет конкретных ограничений, при условии, что такой фермент будет обладать активностью разложения цепи РНК гибрида РНК-ДНК. Так, например, предпочтительно, используется РНКаза H, происходящая от Escherichia coli. Такая РНКаза H, происходящая от Escherichia coli, может присутствовать в виде мономера в реакционном растворе в концентрации в пределах 0,2 нМ - 200 нМ, а предпочтительно, она может присутствовать в этом растворе в концентрации в пределах 0,2 нМ - 200 нМ, 0,2 нМ - 100 нМ, 0,2 нМ - 50 нМ, 1 нМ - 200 нМ, 1 нМ - 100 нМ, 1 нМ - 50 нМ или 10 нМ - 50 нМ, однако, эти концентрации не ограничиваются вышеуказанными пределами.
[0074] Примером третьей группы ферментов, которые катализируют разделение двух сестринских кольцевых ДНК, является группа ферментов, представленная, например, как группа ферментов, описанная Peng H & Marians KJ PNAS 1993, 90: 8571-8575. В частности, примерами третьей группы ферментов являются один или более ферментов, выбранных из группы, состоящей из фермента, обладающего активностью топоизомеразы IV; фермента, обладающего активностью топоизомеразы III; и фермента, обладающего геликазной активностью типа RecQ; или комбинации вышеупомянутых ферментов.
[0075] Биологическое происхождение фермента, обладающего активностью топоизомеразы III, не имеет конкретных ограничений, при условии, что такой фермент будет обладать активностью, аналогичной активности топоизомеразы III Escherichia coli. Так, например, предпочтительно, используется топоизомераза III, происходящая от Escherichia coli. Такая топоизомераза III, происходящая от Escherichia coli, может присутствовать в виде мономера в реакционном растворе в концентрации в пределах 20 нМ - 500 нМ, а предпочтительно, она может присутствовать в этом растворе в концентрации в пределах 20 нМ - 400 нМ, 20 нМ - 300 нМ, 20 нМ - 200 нМ, 20 нМ - 100 нМ, или 30 нМ - 80 нМ, однако, эти концентрации не ограничиваются вышеуказанными пределами.
[0076] Биологическое происхождение фермента, обладающего геликазной активностью типа RecQ, не имеет конкретных ограничений, при условии, что такой фермент будет обладать активностью, аналогичной активности RecQ Escherichia coli. Так, например, предпочтительно, используется RecQ, происходящий от Escherichia coli. Такой RecQ, происходящий от Escherichia coli, может присутствовать в виде мономера в реакционном растворе в концентрации в пределах 20 нМ - 500 нМ, а предпочтительно, он может присутствовать в этом растворе в концентрации в пределах 20 нМ - 400 нМ, 20 нМ - 300 нМ, 20 нМ - 200 нМ, 20 нМ - 100 нМ, или 30 нМ - 80 нМ, однако, эти концентрации не ограничиваются вышеуказанными пределами.
[0077] Биологическое происхождение фермента, обладающего активностью топоизомеразы IV, не имеет конкретных ограничений, при условии, что такой фермент будет обладать активностью, аналогичной активности топоизомеразы IV Escherichia coli. Так, например, предпочтительно, используется топоизомераза IV, происходящая от Escherichia coli и представляющая собой комплекс ParC и ParE. Такая топоизомераза IV, происходящая от Escherichia coli, может присутствовать в виде гетеротетрамера в реакционном растворе в концентрации в пределах 0,1 нМ - 50 нМ, а предпочтительно, она может присутствовать в этом растворе в концентрации в пределах 0,1 нМ - 40 нМ, 0,1 нМ - 30 нМ, 0,1 нМ - 20 нМ, 1 нМ - 40 нМ, 1 нМ - 30 нМ, 1 нМ - 20 нМ, 1 нМ - 10 нМ, или 1 нМ - 5 нМ, однако, эти концентрации не ограничиваются вышеуказанными пределами.
[0078] Первая, вторая и третья группы ферментов, указанных выше, могут представлять собой ферменты, которые являются коммерчески доступными, или могут быть экстрагированы из микроорганизмов и очищены, если это необходимо. Экстракция и очистка ферментов из микроорганизмов могут быть осуществлены, если это необходимо, методами, известными специалистам.
[0079] Если в качестве первой, второй и третьей группы ферментов используются ферменты, отличающиеся от вышеописанных ферментов, происходящих от Escherichia coli, то каждый из этих ферментов может быть использован в концентрации в пределах, соответствующих значений единицы активности фермента, и такая концентрация должна быть аналогична концентрации вышеописанного фермента, происходящего от Escherichia coli.
[0080] В качестве dNTP, содержащегося в реакционной смеси, используемой в методе RCR-амплификации, могут быть использованы dNTP, аналогичные dNTP, используемым в способе получения ДНК согласно изобретению.
[0081] Если это необходимо, то реакционная смесь, полученная методом RCR-амплификации, может также содержать источник ионов магния, источник ионов щелочного металла и АТФ.
[0082] В методе RCR-амплификации, концентрация АТФ, содержащегося в реакционной смеси в начале реакции, может составлять, например, в пределах 0,1-3 мМ, предпочтительно, в пределах 0,1-2 мМ, более предпочтительно, в пределах 0,1-1,5 мМ, а еще более предпочтительно, в пределах 5-1,5 мМ.
[0083] В качестве источника ионов магния, включенного в реакционную смесь, используемую в методе RCR-амплификации, могут быть использованы те же самые вещества, которые используются в способе получения ДНК согласно изобретению. В методе RCR-амплификации, концентрация источника ионов магния, содержащегося в реакционной смеси в начале реакции, может представлять собой, например, концентрацию, необходимую для введения в реакционную смесь 5-50 мМ ионов магния.
[0084] В качестве источника ионов щелочного металла, включенного в реакционную смесь, используемую в методе RCR-амплификации, могут быть использованы те же самые вещества, которые используются в способе получения ДНК согласно изобретению. В методе RCR-амплификации, концентрация источника щелочного металла, содержащегося в реакционной смеси в начале реакции, может представлять собой, например, концентрацию, необходимую для введения в реакционный раствор ионов щелочного металла в пределах 100 мМ или более, а предпочтительно, 100 мМ-300 мМ, однако, эти концентрации не ограничиваются вышеуказанными пределами.
[0085] В методе RCR-амплификации, количество связанной молекулы, содержащейся в реакционной смеси, не имеет конкретных ограничений. Так, например, в начале реакции, связанная молекула может присутствовать в смеси в концентрации 10 нг/мкл или менее, 5 нг/мкл или менее, 1 нг/мкл или менее, 0,8 нг/мкл или менее, 0,5 нг/мкл или менее, или 0,3 нг/мкл или менее.
[0086] При инкубировании полученной реакционной смеси в изотермических условиях при предварительно определенной температуре, амплифицируют только кольцевую ДНК, содержащую последовательность ориджина репликации, способную связываться с ферментом, обладающим активностью DnaA. Температура реакции при RCR-амплификации не имеет конкретных ограничений, при условии, что будет проходить реакция репликации ДНК. Более конкретно, температура может составлять, например, в пределах 20-80°C, 25-50°C или 25-40°C, и такая температура является оптимальной температурой для ДНК-полимеразы. Время реакции при RCR-амплификации может быть соответствующим образом выбрано в зависимости от количества продукта амплификации кольцевой связанной молекулы-мишени. Время реакции может составлять, например, от 30 минут до 24 часов.
[0087] RCR-амплификация может быть также осуществлена путем инкубирования полученной реакционной смеси в соответствии с термоциклом, который повторяется при инкубировании при 30°C или выше и при инкубировании при 27°C или ниже. Инкубирование при 30°C или выше не имеет конкретных ограничений, при условии, что такая температура представляет собой температуру в пределах, достаточных для инициации репликации кольцевой ДНК, включающей оriС. Так, например, температура может составлять 30-80°С, 30-50°С, 30-40°С или 37°C. Инкубирование при 30°C или выше может быть осуществлено в течение периода времени от 10 секунд до 10 минут за один цикл, однако, этот период времени не имеет конкретных ограничений. Инкубирование при 27°C или ниже не имеет конкретных ограничений, при условии, что оно будет осуществлено при температуре, при которой будет подавляться инициация репликации и будет проходить реакция элонгации ДНК. Так, например, температура может составлять 10-27°С, 16-25°С или 24°C. Инкубирование при 27°C или ниже может быть, предпочтительно, проведено в зависимости от длини амплифицируемой кольцевой ДНК, но не имеет конкретных ограничений. Так, например, инкубирование может быть осуществлено в течение 1-10 секунд на 1000 оснований за один цикл. Число термоциклов не имеет конкретных ограничений, но оно может составлять от 10 до 50 циклов, от 20 до 40 циклов, от 25 до 35 циклов или 30 циклов.
[0088] Связанную молекулу, полученную посредством реакции связывания, перед ее использованием в реакции репарации гэпов и ников и для RCR-амплификации в качестве матрицы, предпочтительно, подвергают термоинкубированию при 50°C-70°C с последующим быстрым охлаждением. Время проведения термообработки не имеет конкретных ограничений и может составлять, например, 1-15 минут, а предпочтительно, 2-10 минут. Температура быстрого охлаждения не имеет конкретных ограничений и составляет, например, 10°C или ниже, а предпочтительно, 4°C или ниже. Скорость охлаждения составляет предпочтительно 50°C/минуту или более, более предпочтительно 70°C/минуту или более, а еще более предпочтительно 85°C/минуту или более. Так, например, реакционная смесь в контейнере может быть подвергнута быстрому охлаждению после термообработки путем ее помещения непосредственно на лед или контактирования с металлическим блоком, доведенным до 4°C или менее.
[0089] Реакционный раствор, полученный сразу после реакции связывания, содержит связанные молекулы, полученные посредством неспецифического связывания. Неспецифическое связывание может быть подвергнуто диссоциации посредством термообработки и быстрого охлаждения реакционного раствора. По этой причине, реакции репарации гэпов и ников и реакции RCR-амплификации осуществляют с использованием связанной молекулы в качестве матрицы после термообработки и быстрого охлаждения. Это приводит к подавлению неспецифического связывания, в результате чего может быть эффективно получена полноразмерная двухцепочечная ДНК представляющей интерес связанной молекулы.
[0090] В способе получения ДНК согласно изобретению, амплификацию линейной или кольцевой связанной молекулы, полученной посредством реакции связывания, осуществляют в микроорганизме путем введения в него связанной молекулы. Это может быть осуществлено с использованием фермента этого микроорганизма или т.п. Связанная молекула, вводимая в микроорганизм, может представлять собой связанную молекулу до реакции репарации гэпов и ников или связанную молекулу после реакции репарации. Даже при непосредственном введении в микроорганизм связанной молекулы, имеющей гэп и ник, связанная молекула в полноразмерной двухцепочечной ДНК без гэпов и ников может быть получена в виде продукта амплификации. Примерами микроорганизмов, в которые вводят связанную молекулу, являются Escherichia coli, Bacillus subtilis, актиномицеты, архебактерии, дрожжи, нитчатые грибы и т.п. Введение связанной молекулы в микроорганизм может быть осуществлено стандартным методом, таким как метод электропорации. Амплифицированная связанная молекула может быть выделена из микроорганизма стандартным методом.
[0091] Набор для связывания фрагментов ДНК
Набор для связывания фрагментов ДНК согласно изобретению представляет собой набор для связывания фрагментов ДНК двух или более типов друг с другом в областях, где последовательности оснований являются гомологичными, с получением линейной или кольцевой ДНК, и содержит белок рекомбиназу семейства RecA. Набор, если он используется для связывания линейных двухцепочечных фрагментов ДНК, также, предпочтительно, содержит экзонуклеазу. Белок рекомбиназу семейства RecA и экзонуклеазу, содержащиеся в наборе, добавляют к раствору, содержащему связанные фрагменты ДНК двух или более типов. Таким образом, способ получения ДНК согласно изобретению может быть осуществлен более простым методом, и в этом случае, может быть легко получена представляющая интерес связанная молекула. Поскольку в наборе содержится белок рекомбиназы семейства RecA, то могут быть использованы ферменты, аналогичные ферментам, используемым в способе получения ДНК согласно изобретению. Экзонуклеаза, содержащаяся в наборе, предпочтительно, представляет собой 3'→5'-экзонуклеазу. Более предпочтительно, набор содержит 3'→5'-экзонуклеазу, специфичную к линейной двухцепочечной ДНК, а еще более предпочтительно, 3'→5'-экзонуклеазу, специфичную к линейной двухцепочечной ДНК, и 3'→5'-экзонуклеазу, специфичную к одноцепочечной ДНК.
[0092] Набор для связывания фрагментов ДНК согласно изобретению также предпочтительно включает регенерирующий фермент для нуклеозид-трифосфата или дезоксинуклеотид-трифосфата и его субстрат. Набор для связывания фрагментов ДНК согласно изобретению может включать одно или более веществ, выбранных из группы, состоящей из нуклеозид-трифосфата, дезоксинуклеотид-трифосфата, источника ионов магния, источника ионов щелочного металла, диметилсульфоксида, хлорида тетраметиламмония, полиэтиленгликоля, дитиотреитола и буфера. Любые из этих веществ, используемых в способе получения ДНК согласно изобретению, могут быть использованы в форме, указанной выше.
[0093] Набор для связывания фрагментов ДНК согласно изобретению также предпочтительно включает документ, в котором описан протокол осуществления способа получения ДНК согласно изобретению с использованием данного набора. Этот протокол может быть отпечатан на поверхности контейнера, содержащего этот набор.
Примеры
[0094] Настоящее изобретение более подробно описано ниже со ссылкой на Примеры, которые не рассматриваются как ограничение объема изобретения.
[0095] [Пример 1]
Линейные двухцепочечные фрагменты ДНК двух или более типов были присоединены с использованием белка рекомбиназы семейства RecA и 3'→5'-экзонуклеазы. В этой реакции было исследовано влияние концентрации источника ионов магния и АТФ в реакционном растворе.
[0096] DCW1-DCW7 (SEQ ID NO: 1 - SEQ ID NO: 7), которые представляют собой линейные двухцепочечные фрагменты ДНК размером 591 п.о., были использованы в виде связанных линейных двухцепочечных фрагментов ДНК. Область от конца до 60 оснований каждого линейного двухцепочечного фрагмента ДНК представляет собой гомологичную область. То есть, область в 60 оснований от 532-го до 591-го основания в DCW1 представляет собой гомологичную область для связывания с DCW2 и состоит из той же самой последовательности оснований, как и последовательность из 60 оснований от 1-го до 60-го основания в DCW2. Область в 60 оснований от 532-го до 591-го основания в DCW2 представляет собой гомологичную область для связывания с DCW3 и состоит из той же самой последовательности оснований, как и последовательность из 60 оснований от 1-го до 60-го основания в DCW3. Область в 60 оснований от 532-го до 591-го основания в DCW3 представляет собой гомологичную область для связывания с DCW4 и состоит из той же самой последовательности оснований, как и последовательность из 60 оснований от 1-го до 60-го основания в DCW4. Область в 60 оснований от 532-го до 591-го основания в DCW4 представляет собой гомологичную область для связывания с DCW5 и состоит из той же самой последовательности оснований, как и последовательность из 60 оснований от 1-го до 60-го основания в DCW5. Область в 60 оснований от 532-го до 591-го основания в DCW5 представляет собой гомологичную область для связывания с DCW6 и состоит из той же самой последовательности оснований, как и последовательность из 60 оснований от 1-го до 60-го основания в DCW6. Область в 60 оснований от 532-го до 591-го основания в DCW6 представляет собой гомологичную область для связывания с DCW7 и состоит из той же самой последовательности оснований, как и последовательность из 60 оснований от 1-го до 60-го основания в DCW7.
[0097] В качестве белка рекомбиназы семейства RecA использовали RecA E. coli дикого типа (SEQ ID NO: 61), а в качестве 3'→5'-экзонуклеазы использовали экзонуклеазу III.
[0098] В частности, сначала приготавливали реакционные растворы, состоящие из 1 нМ каждого из DCW1-DCW7, 1 мкМ RecA, 40 мЕд/мкз экзонуклеазы III, 20 мМ Триса-HCl (рН 8,0), 4 мМ DТТ, 1 мМ или 10 мМ ацетата магния, 30 мкМ, 100 мкМ, 300 мкМ или 1000 мкМ АТФ, 4 мМ фосфата креатина, 20 нг/мкл креатинкиназы, 50 мМ глутамата калия, 150 мМ ТМАС, 5% по массе ПЭГ8000 и 10% по объему ДМСО. Затем эти реакционные растворы инкубировали при 42°C в течение 2 часов для проведения реакции связывания. 1 мкл реакционного раствора после завершения реакции подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR (зарегистрированный торговый знак).
[0099] Результаты окрашивания представлены на фиг. 2. На этой фигуре «500 п.о.» означает дорожку, где был использован маркер ДНК-лэддера, состоящий из полос 500 п.о. - 5 т.п.о. (всего 10) с интервалами в 500 п.о., а «Вход» означает дорожку, где используется 1 мкл раствора, содержащего 1 нМ каждого из DCW1-DCW7. На этой фигуре, «7 frag» обозначает полосу связанной молекулы, в которой были связаны все семь фрагментов DCW1-DCW7.
[0100] Из реакционных растворов с 1 мМ ацетата магния, в реакционном растворе с 30 мкМ АТФ, связанная молекула почти не детектировалась, а в реакционных растворах со 100 мкМ, 300 мкМ и 1000 мкМ АТФ детектировались полосы связанных молекул 6 типов, которые простирались от связанной молекулы из 2 фрагментов до связанной молекулы из 7 фрагментов. При сравнении результатов, полученных для реакционных растворов со 100 мкМ, 300 мкМ и 1000 мкМ АТФ, было обнаружено, что реакционный раствор со 100 мкМ АТФ содержал наибольшее количество связанной молекулы, полученной путем связывания фрагментов всех 7 типов, а полосы несвязанных фрагментов не детектировались. В противоположность этому, в реакционном растворе с 1000 мкМ АТФ, полоса связанной молекулы из 7 фрагментов была очень слабой, и многие фрагменты оставались несвязанными.
С другой стороны, в реакционных растворах с 10 мМ ацетата магния, полосы связанных молекул 6 типов от связанной молекулы из 2 фрагментов до связанной молекулы из 7 фрагментов детектировались в реакционных растворах с 30 мкМ и 100 мкМ АТФ. В реакционных растворах с 300 мкМ и 1000 мкМ АТФ детектировались полосы связанных молекул от связанной молекулы из 2 фрагментов до связанной молекулы из 4 фрагментов, но при этом, не детектировались полосы связанных молекул со связанными 5 или более фрагментами, и многие фрагменты оставались несвязанными.
Из всех образцов, количество полученной связанной молекулы из 7 фрагментов было наибольшим в реакционном растворе с 1 мМ ацетата магния и 100 мМ АТФ. Эти результаты позволяют сделать следующие выводы: для связывания множества фрагментов важную роль играет баланс между концентрацией ионов магния и АТФ в реакционном растворе. А в случае, если концентрация АТФ является слишком высокой, то это может приводить к ингибированию реакции связывания.
[0101] [Пример 2]
Линейные двухцепочечные фрагменты ДНК двух или более типов были присоединены с использованием белка рекомбиназы семейства RecA и 3'→5'-экзонуклеазы. В этой реакции было исследовано влияние концентрации ПЭГ 8000 и АТФ-регенерирующей системы в реакционном растворе.
[0102] Lter1 - Lter5 (SEQ ID NO: 54 - SEQ ID NO: 58), которые представляют собой линейные двухцепочечные фрагменты ДНК размером 3100 п.о. или 2650 п.о., были использованы в виде связанных линейных двухцепочечных фрагментов ДНК. Область от конца до 60 оснований каждого линейного двухцепочечного фрагмента ДНК представляет собой гомологичную область. То есть, область в 60 оснований от 3041-го до 3100-го основания в Lter1 представляет собой гомологичную область для связывания с Lter2 и состоит из такой же последовательности оснований, как и последовательность из 60 оснований от 1-го до 60-го основания в Lter2. Область в 60 оснований от 3041-го до 3100-го основания в Lter2 представляет собой гомологичную область для связывания с Lter3 и состоит из такой же последовательности оснований, как и последовательность из 60 оснований от 1-го до 60-го основания в Lter3. Область в 60 оснований от 3041-го до 3100-го основания в Lter3 представляет собой гомологичную область для связывания с Lter4 и состоит из такой же последовательности оснований, как и последовательность из 60 оснований от 1-го до 60-го основания в Lter4. Область в 60 оснований от 3041-го до 3100-го основания в Lter4 представляет собой гомологичную область для связывания с Lter5 и состоит из такой же последовательности оснований, как и последовательность из 60 оснований от 1-го до 60-го основания в Lter5.
[0103] В качестве белка рекомбиназы семейства RecA использовали мутант F203W RecA E. coli, а в качестве 3'→5'-экзонуклеазы использовали экзонуклеазу III. В качестве АТФ-регенерирующего фермента использовали креатинкиназу, а в качестве субстрата использовали фосфат креатина.
[0104] В частности, сначала приготавливали реакционные растворы, состоящие из 0,03 нМ каждого Lter1-Lter5, 1 мкМ мутанта F203W RecA, 40 мЕд/мкл экзонуклеазы III, 20 мМ Триса-HCl (рН 8,0), 10 мМ ацетата магния, 100 мкМ АТФ, 4 мМ фосфата креатина, 20 нг/мкл креатинкиназы, 150 мМ ацетата калия и 0%, 2%, 5% или 10% по массе ПЭГ8000. При этом отдельно приготавливали реакционные растворы аналогичным способом, за исключением того, что они не содержали фосфата креатина и креатинкиназы. Затем эти реакционные растворы инкубировали при 30°C в течение 30 минут для проведения реакции связывания. 4 мкл реакционного раствора после завершения реакции подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0105] Результаты окрашивания представлены на фиг. 3. На этой фигуре «5 frag» означает полосу связанной молекулы, в которой все пять фрагментов Lter1-Lter5 были связаны. Среди реакционных растворов, не содержащих фосфата креатина и креатинкиназы, в реакционном растворе с 0% по массе ПЭГ8000 детектировались полосы связанной молекулы из 2 фрагментов и связанной молекулы из 3 фрагментов, а полосы связанных молекул с 4 или более связанными фрагментами не детектировались. В противоположность этому, полосы связанной молекулы из 4 фрагментов и связанной молекулы из 5 фрагментов также детектировались в реакционном растворе с высокой концентрацией ПЭГ8000. С другой стороны, среди реакционных растворов, содержащих фосфат креатина и креатинкиназу, полосы связанной молекулы из 2 фрагментов, связанной молекулы из 3 фрагментов и связанной молекулы из 4 фрагментов детектировались в реакционных растворах с 0% по массе ПЭГ8000, а полоса связанной молекулы из 5 фрагментов не детектировалась. В противоположность этому, полоса связанной молекулы из 5 фрагментов также детектировались в реакционном растворе с высокой концентрацией ПЭГ8000. В результате сравнения реакционных растворов с 0% по массе ПЭГ8000 было обнаружено, что реакционный раствор с АТФ-регенерирующей системой с большей вероятностью будет образовывать связанную молекулу из множества фрагментов. В реакционном растворе без фосфата креатина и креатинкиназы и в реакционном растворе, содержащем эти соединения, количество продуцируемой связанной молекулы из 5 фрагментов в реакционном растворе с ПЭГ превышало количество этой молекулы в реакционном растворе без ПЭГ. Исходя их этих результатов было обнаружено, что ПЭГ стимулирует связывание. Из всех этих образцов, реакционный раствор с фосфатом креатина, с креатинкиназой и 5% по массе ПЭГ8000 имели меньшее количество несвязанных фрагментов, и наибольшее количество связанной молекулы из 5 фрагментов.
[0106] [Пример 3]
Линейные двухцепочечные фрагменты ДНК двух или более типов были присоединены с использованием белка рекомбиназы семейства RecA и 3'→5'-экзонуклеазы. В этой реакции было исследовано влияние концентрации ДМСО в реакционном растворе.
[0107] DCW1-DCW5 (SEQ ID NO: 1 - SEQ ID NO: 5) были использованы в качестве связанных линейных двухцепочечных фрагментов ДНК. В качестве белка рекомбиназы семейства RecA использовали мутант F203W RecA E. coli, а в качестве 3'→5'-экзонуклеазы использовали экзонуклеазу III.
[0108] В частности, сначала приготавливали реакционные растворы, состоящие из 1 нМ каждого из DCW1-DCW5, 1 мкМ мутанта F203W RecA, 40 мЕд/мкл экзонуклеазы III, 20 мМ Триса-HCl (рН 8,0), 4 мМ DТТ, 10 мМ ацетата магния, 100 мкМ АТФ, 150 мМ ацетата калия, 5% по массе ПЭГ8000, 0%, 1%, 3% или 10% по объему ДМСО. Затем эти реакционные растворы инкубировали при 30°C в течение 30 минут для проведения реакции связывания. 2 мкл реакционного раствора после завершения реакции подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0109] Результаты окрашивания представлены на фиг. 4(а). На этой фигуре, «МК3» означает дорожку, где был использован маркер ДНК-лэддера, а «Вход» означает дорожку, где используется 2 мкл раствора, содержащего 1 нМ каждого из DCW1-DCW5. В результате, полоса связанной молекулы, в которой были связаны все пять фрагментов, детектировалась во всех образцах после реакции связывания. Реакционный раствор с 10% по объему ДМСО имел большее количество связанной молекулы из 5 фрагментов, чем другие реакционные растворы.
[0110] Затем реакционные растворы приготавливали аналогичным способом, за исключением того, что концентрация ДМСО составляла 0% по объему, 10% по объему, 20% по объему или 40% по объему, и эти реакционные растворы инкубировали при 30°C в течение 30 минут для проведения реакции связывания. 2 мкл реакционного раствора после завершения реакции подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0111] Результаты окрашивания представлены на фиг. 4(b). На этой фигуре, «500 п.о.-лэддер» означает дорожку, где был использован маркер ДНК-лэддера в Примере 1, а «Вход» означает такую же дорожку, как и на фиг. 4(а). В результате уровень продуцирования связанной молекулы, в которой все пять фрагментов были связаны друг с другом, заметно увеличивался в реакционном растворе с 10% по объему или 20% по объему ДМСО, но эта полоса не детектировалась, а связывание ингибировалось в реакционном растворе с 40% по объему ДМСО. На основании этих результатов были сделаны следующие выводы. Реакция связывания стимулировалась в случае, если содержание ДМСО составляло 5% по объему или более. В противоположность этому, если концентрация ДМСО была слишком высокой то реакция связывания ингибировалась.
[0112] [Пример 4]
Линейные двухцепочечные фрагменты ДНК двух или более типов были присоединены с использованием белка рекомбиназы семейства RecA и 3'→5'-экзонуклеазы. В этой реакции было исследовано влияние концентрации ТМАС в реакционном растворе.
[0113] DCW1-DCW7 (SEQ ID NO: 1 - SEQ ID NO: 7) были использованы в виде связанных линейных двухцепочечных фрагментов ДНК. В качестве белка рекомбиназы семейства RecA использовали мутант F203W RecA E. coli, а в качестве 3'→5'-экзонуклеазы использовали экзонуклеазу III.
[0114] В частности, сначала приготавливали реакционные растворы, состоящие из 1 нМ каждого из DCW1-DCW7, 1 мкМ мутанта F203W RecA, 40 мЕд/мкл экзонуклеазы III, 20 мМ Триса-HCl (рН 8,0), 4 мМ DТТ, 10 мМ ацетата магния, 100 мкМ АТФ, 4 мМ фосфата креатина, 20 нг/мкл креатинкиназы, 150 мМ глутамата калия, 5% по массе ПЭГ8000, 10% по объему ДМСО и 0 мМ, 15 мМ, 30 мМ, 60 мМ или 100 мМ ТМАС. Затем эти реакционные растворы инкубировали при 37°C в течение 2 часов для проведения реакции связывания. 1,5 мкл реакционного раствора после завершения реакции подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0115] Результаты окрашивания представлены на фиг. 5(а). На этой фигуре, «500 п.о.-лэддер» означает дорожку, где был использован маркер ДНК-лэддера в Примере 1, а «Вход» означает дорожку, где используется 1 мкл раствора, содержащего 1 нМ каждого из DCW1-DCW7. В результате, полоса связанной молекулы, в которой были связаны все семь фрагментов, детектировалась во всех образцах после реакции связывания. В частности, количество связанной молекулы из 7 фрагментов в реакционном растворе с 100 мМ ТМАС заметно превышало количество, наблюдаемое в других реакционных растворах.
[0116] Затем, реакционные растворы приготавливали аналогичным способом, за исключением того, что концентрация ТМАС составляла 60 мМ, 100 мМ, 150 мМ, 200 мМ или 250 мМ, а концентрация глутамата калия составляла 50 мМ. Эти реакционные растворы инкубировали при 42°C в течение 2 часов для проведения реакции связывания. 1,9 мкл реакционного раствора после завершения реакции подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0117] Результаты окрашивания представлены на фиг. 5(b). В результате, реакционные растворы c 100 мМ - 200 мМ ТМАС имели явно большее содержание связанной молекулы из 7 фрагментов, чем реакционный раствор с 60 мМ ТМАС, и их эффективность связывания повышалась. Реакционный раствор со 150 мМ ТМАС имел наибольшее количество связанной молекулы из 7 фрагментов. С другой стороны, в реакционном растворе с 250 мМ ТМАС количество связанной молекулы из 7 фрагментов было меньше, чем в реакционном растворе с 60 мМ ТМАС, и оставалось множество несвязанных фрагментов. Исходя из этого результата был сделан вывод, что связывание стимулируется ТМАС в количестве 100-200 мМ.
[0118] [Пример 5]
Линейные двухцепочечные фрагменты ДНК двух или более типов были присоединены с использованием белка рекомбиназы семейства RecA и 3'→5'-экзонуклеазы. В этой реакции было исследовано влияние источников ионов щелочного металла в реакционном растворе.
[0119] DCW1-DCW7 или DCW1-DCW5 были использованы в качестве виде связанных линейных двухцепочечных фрагментов ДНК. В качестве белка рекомбиназы семейства RecA использовали мутант F203W RecA E. coli, а в качестве 3'→5'-экзонуклеазы использовали экзонуклеазу III.
[0120] В частности, сначала приготавливали реакционные растворы, состоящие из 1 нМ каждого из DCW1-DCW5, 1 мкМ мутанта F203W RecA, 40 мЕд/мкл экзонуклеазы III, 20 мМ Триса-HCl (рН 8,0), 4 мМ DТТ, 10 мМ ацетата магния, 100 мкМ АТФ, 4 мМ фосфата креатина, 20 нг/мкл креатинкиназы, 0 мМ, 50 мМ, 75 мМ, 100 мМ, 125 мМ или 150 мМ ацетата калия, 5% по массе ПЭГ8000, и 10% по объему ДМСО. Эти реакционные растворы получали аналогичным образом, за исключением того, что вместо ацетата калия они содержали 150 мМ глутамата калия. Затем реакционные растворы инкубировали при 30°C в течение 2 часов для проведения реакции связывания. 2 мкл реакционного раствора после завершения реакции подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0121] Результаты окрашивания представлены на фиг. 6. Полоса связанной молекулы, в которой были связаны все пять фрагментов, не детектировалась в реакционных растворах, не содержащих источников ионов щелочного металла, и детектировалась во всех реакционных растворах, содержащих ацетат калия или глутамат калия. В частности, полоса несвязанных фрагментов в реакционном растворе с глутаматом калия была слабее, чем в реакционном растворе с ацетатом калия. Таким образом, было высказано предположение, что глутамат калия может значительно повышать эффективность связывания по сравнению с ацетатом калия.
[0122] Затем, реакционные растворы приготавливали аналогичным способом, за исключением того, что в качестве связанных линейных двухцепочечных фрагментов ДНК были использованы DCW1-DCW7, и концентрация каждого из них составляла 1 нМ, в качестве источника ионов щелочного металла использовали глутамат калия, а концентрация глутамата калия составляла 50 мМ или 150 мМ. Эти реакционные растворы инкубировали при 37°C, 42°C или 45°C в течение 1 часа для проведения реакции связывания. 1 мкл реакционного раствора после завершения реакции подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0123] Результаты окрашивания представлены на фиг. 7. На этой фигуре, «500 п.о.-лэддер» означает дорожку, где был использован маркер ДНК-лэддера в Примере 1, а «Вход» означает дорожку, где используется 1 мкл раствора, содержащего 1 нМ каждого из DCW1-DCW7. В результате, при температурах реакции 37°C и 42°C, реакционные растворы с 50 мМ глутамата калия обнаруживали большую эффективность связывания, чем реакционный раствор со 150 мМ глутамата калия, и была детектирована полоса связанной молекулы, в которой все семь фрагментов были связанными. Исходя из этих результатов было обнаружено, что реакция связывания может ингибироваться в том случае, если концентрация глутамата калия является слишком высокой. В реакционных растворах, инкубированных при 45°C, полоса связанной молекулы из 7 фрагментов не детектировалась даже, если концентрация глутамата калия составляла 50 мМ. Количество связанной молекулы из 7 фрагментов было наибольшим в реакционном растворе, который содержал 50 мМ глутамата калия и был инкубирован при 42°C.
[0124] [Пример 6]
Линейные двухцепочечные фрагменты ДНК двух или более типов были связаны с использованием белка рекомбиназы семейства RecA и 3'→5'-экзонуклеазы. В этой реакции было исследовано влияние молярного отношения каждого линейного двухцепочечного фрагмента ДНК в реакционном растворе.
[0125] DCW1-DCW7 были использованы в виде связанных линейных двухцепочечных фрагментов ДНК. В качестве белка рекомбиназы семейства RecA использовали мутант F203W RecA E. coli, а в качестве 3'→5'-экзонуклеазы использовали экзонуклеазу III.
[0126] В частности, сначала приготавливали реакционные растворы, состоящие из 1 нМ каждого из DCW1-DCW7, 1 мкМ мутанта F203W RecA, 40 мЕд/мкл экзонуклеазы III, 20 мМ Триса-HCl (рН 8,0), 4 мМ DТТ, 10 мМ ацетата магния, 100 мкМ АТФ, 4 мМ фосфата креатина, 20 нг/мкл креатинкиназы, 50 мМ ацетата калия, 5% по массе ПЭГ8000, и 10% по объему ДМСО. Эти реакционные растворы получали аналогичным образом, за исключением того, что концентрация DCW3 составляла 2 нМ. Затем реакционные растворы инкубировали при 42°C в течение 2 часов для проведения реакции связывания. 1 мкл реакционного раствора после завершения реакции подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0127] Результаты окрашивания представлены на фиг. 8. На этой фигуре, «500 п.о.-лэддер» означает дорожку, где был использован маркер ДНК-лэддера в Примере 1, а «Вход» означает дорожку, где используется 1 мкл раствора, содержащего 1 нМ каждого из DCW1-DCW7. Термин «равные» означает дорожку, где используется реакционный раствор, содержащий 1 нМ каждого из DCW1 ~ DCW7. «2-кратный избыток 3-го фрагмента» означает дорожку, где присутствует реакционный раствор, содержащий 1 нМ каждого из DCW1-DCW7, за исключением DCW3, концентрация которого составляла 2 нМ DCW3. В результате, во всем реакционном растворе была получена связанная молекула из 7 фрагментов. Однако, в реакционном растворе, содержащем только двойную концентрацию (два моля) DCW3, количество связанной молекулы из 7 фрагментов снижалось, а количество связанной молекулы из 3 фрагментов и количество связанной молекулы из 5 фрагментов повышалось. Было высказано предположение, что избыточное количество DCW3 приводило к повышению числа связанных молекул, в которых DCW1-DCW3 были связаны, и числа связанных молекул, в которых DCW3-DCW7 были связаны. Исходя из этих результатов было обнаружено, что эффективность связывания множества фрагментов была повышена после получения реакционного раствора, где молярные отношения каждого связанного фрагмента были одинаковыми.
[0128] [Пример 7]
Линейные двухцепочечные фрагменты ДНК двух или более типов были связаны с использованием белка рекомбиназы семейства RecA и 3'→5'-экзонуклеазы. В этой реакции было исследовано влияние концентрации 3'→5'-экзонуклеазы и времени проведения реакции в реакционном растворе.
[0129] DCW1-DCW7 были использованы в виде связанных линейных двухцепочечных фрагментов ДНК. В качестве белка рекомбиназы семейства RecA использовали RecA E. coli дикого типа, а в качестве 3'→5'-экзонуклеазы использовали экзонуклеазу III.
[0130] В частности, сначала приготавливали реакционные растворы, состоящие из 1 нМ каждого из DCW1-DCW7, 1 мкМ RecA E. coli дикого типа, 20 мЕд/мкл, 40 мЕд/мкл, 80 мЕд/мкл, 120 мЕд/мкл или 160 мЕд/мкл экзонуклеазы III, 20 мМ Триса-HCl (рН 8,0), 4 мМ DТТ, 1 мМ ацетата магния, 100 мкМ АТФ, 4 мМ фосфата креатина, 20 нг/мкл креатинкиназы, 50 мМ глутамата калия, 150 мМ ТМАС, 5% по массе ПЭГ8000 и 10% по объему ДМСО. Затем реакционные растворы инкубировали при 42°C в течение 15 минут, 30 минут или 60 минут для проведения реакции связывания. 1 мкл реакционного раствора после завершения реакции подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0131] Результаты окрашивания представлены на фиг. 9. На этой фигуре, «500 п.о.-лэддер» означает дорожку, где был использован маркер ДНК-лэддера в Примере 1, а «Вход» означает дорожку, где используется 1 мкл раствора, содержащего 1 нМ каждого из DCW1-DCW7. В результате, в реакционных растворах с 20 мЕд/мкл, связанная молекула почти не образовывалась даже за время инкубирования 60 минут. В противоположность этому, в реакционных растворах с 40 мЕд/мкл экзонуклеазы III, связанная молекула почти не образовывалась за время инкубирования 30 минут, но связанная молекула из 7 фрагментов образовывалась за время инкубирования 60 минут. В реакционных растворах с 80-160 мЕд/мкл экзонуклеазы III, связанная молекула из 7 фрагментов образовывалась даже за время инкубирования 15 минут. Реакционный раствор, содержащий 80 мЕд/мкл экзонуклеазы III и инкубируемый в течение 30 минут, имел наибольшее количество связанной молекулы из 7 фрагментов и обнаруживал наилучшую эффективность связывания множества фрагментов.
[0132] [Пример 8]
Линейные двухцепочечные фрагменты ДНК двух или более типов были связаны с использованием белка рекомбиназы семейства RecA и 3'→5'-экзонуклеазы. В этой реакции было исследовано влияние концентрации линейного двухцепочечного фрагмента ДНК в реакционном растворе.
[0133] DCW1-DCW49 (SEQ ID NО: 1 - SEQ ID NО: 49) были использованы в виде связанных линейных двухцепочечных фрагментов ДНК. По аналогии с DCW1-DCW7, области DCW8-DCW49, простирающиеся от каждого конца до 60 оснований, представляют собой гомологичные области. В качестве белка рекомбиназы семейства RecA использовали RecA E. coli дикого типа, а в качестве 3'→5'-экзонуклеазы использовали экзонуклеазу III.
[0134] В частности, сначала приготавливали реакционные растворы, состоящие из 1 нМ или 0,5 нМ каждого из линейных двухцепочечных фрагментов ДНК, 1 мкМ RecA E. coli дикого типа, 80 мЕд/мкл экзонуклеазы III, 20 мМ Триса-HCl (рН 8,0), 4 мМ DТТ, 1 мМ ацетата магния, 100 мкМ АТФ, 4 мМ фосфата креатина, 20 нг/мкл креатинкиназы, 50 мМ глутамата калия, 150 мМ ТМАС, 5% по массе ПЭГ8000 и 10% по объему ДМСО. В реакционном растворе, содержащем 1 нМ каждого из DCW1 - DCW20, общее количество линейных двухцепочечных фрагментов ДНК составляет 20 нМ (7,8 нг/мкл). В реакционном растворе, содержащем 1 нМ каждого из DCW1 - DCW25, общее количество линейных двухцепочечных фрагментов ДНК составляет 25 нМ (9,8 нг/мкл). В реакционном растворе, содержащем 1 нМ каждого из DCW1 - DCW30, общее количество линейных двухцепочечных фрагментов ДНК составляет 30 нМ (11,7 нг/мкл). В реакционном растворе, содержащем 1 нМ каждого из DCW1 - DCW40, общее количество линейных двухцепочечных фрагментов ДНК составляет 40 нМ (15,6 нг/мкл). В реакционном растворе, содержащем 1 нМ каждого из DCW1 - DCW49, общее количество линейных двухцепочечных фрагментов ДНК составляет 49 нМ (19,1 нг/мкл). В реакционном растворе, содержащем 0,5 нМ каждого из DCW1 - DCW49, общее количество линейных двухцепочечных фрагментов ДНК составляет 24,5 нМ (9,6 нг/мкл). Затем, эти реакционные растворы инкубировали при 42°C в течение 30 минут для проведения реакции связывания. Реакционный раствор в нижеуказанных объемах после завершения реакции подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR. Объем реакционного раствора, полученного с использованием 1 нМ каждого из DCW1 - DCW20, составлял 1,25 мкл; объем реакционного раствора, полученного с использованием 1 нМ каждого из DCW1 - DCW25, составлял 1 мкл; объем реакционного раствора, полученного с использованием 1 нМ каждого из DCW1 - DCW30, составлял 0,83 мкл; объем реакционного раствора, полученного с использованием 1 нМ каждого из DCW1 - DCW40, составлял 0,63 мкл; объем реакционного раствора, полученного с использованием 1 нМ каждого из DCW1 - DCW49, составлял 0,51 мкл; и объем реакционного раствора, полученного с использованием 0,5 нМ каждого из DCW1 - DCW49, составлял 1,02 мкл.
[0135] Результаты окрашивания представлены на фиг. 10. В реакционных растворах, содержащих 1 нМ каждого из линейных двухцепочечных фрагментов ДНК, образование связанной молекулы из множества фрагментов затрудняется, поскольку повышается число содержащихся фрагментов, то есть, повышается общее количество линейных двухцепочечных фрагментов ДНК в реакционном растворе. По сравнению с реакционными растворами, содерщими DCW1 - DCW49, связанные молекулы, которые были связаны с большим числом фрагментов, образовывались в реакционном растворе, содержащем 0,5 нМ каждого из линейных двухцепочечных фрагментов ДНК, но не в реакционном растворе, содержащем 1 нМ каждого из линейных двухцепочечных фрагментов ДНК. Эти результаты позволяют предположить, что эффективность связывания может подавляться из-за очень большого общего количества линейных двухцепочечных фрагментов ДНК в реакционном растворе.
[0136] [Пример 9]
Линейные двухцепочечные фрагменты ДНК двух или более типов были связаны с использованием белка рекомбиназы семейства RecA и 3'→5'-экзонуклеазы. В этой реакции было исследовано влияние типов белка рекомбиназы семейства RecA в реакционном растворе.
[0137] DCW1-DCW25 (SEQ ID NO: 1 - SEQ ID NO: 25) были использованы в виде связанных линейных двухцепочечных фрагментов ДНК. В качестве белка рекомбиназы семейства RecA использовали RecA E. coli дикого типа или его мутант F203W, а в качестве 3'→5'-экзонуклеазы использовали экзонуклеазу III.
[0138] В частности, сначала приготавливали реакционные растворы, состоящие из 1 нМ каждого из DCW1-DCW25, 0,5 мкМ, 0,75 мкМ, 1 мкМ, 1,25 мкМ или 1,5 мкМ RecA E. coli дикого типа или его мутанта F203W, 80 мЕд/мкл экзонуклеазы III, 20 мМ Триса-HCl (рН 8,0), 4 мМ DТТ, 1 мМ ацетата магния, 100 мкМ АТФ, 4 мМ фосфата креатина, 20 нг/мкл креатинкиназы, 50 мМ глутамата калия, 150 мМ ТМАС, 5% по массе ПЭГ8000 и 10% по объему ДМСО. Затем реакционные растворы инкубировали при 42°C в течение 30 минут для проведения реакции связывания, а затем инкубировали при 65°C в течение 20 минут. После инкубирования при 65°C и быстрого охлаждения на льду, 1,5 мкл реакционного раствора после завершения реакции подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0139] Результаты окрашивания представлены на фиг. 11. На этой фигуре, «500 п.о.-лэддер» означает дорожку, где был использован маркер ДНК-лэддера в Примере 1, а «Вход» означает дорожку, где используется 1,5 мкл раствора, содержащего 1 нМ каждого из DCW1-DCW25. В результате, независимо от того, является ли белок рекомбиназы семейства RecA белком дикого типа или мутантом F203W, количество продуцируемых связанных молекул из множества фрагментов увеличивалось в зависимости от содержания RecA. Реакционный раствор с мутантом F203W имел большее количество продуцируемых связанных молекул из множества фрагментов и обладал более высокой эффективностью связывания, чем реакционный раствор, содержащий RecA дикого типа.
[0140] [Пример 10]
Линейные двухцепочечные фрагменты ДНК двух или более типов были связаны с использованием белка рекомбиназы семейства RecA и 3'→5'-экзонуклеазы с образованием связанной кольцевой молекулы, и эта связанная кольцевая молекула была подвергнута RCR-амплификации.
[0141] Сначала, в качестве связанных линейных двухцепочечных фрагментов ДНК использовали набор из DCW1-DCW20 (SEQ ID NO: 1 - SEQ ID NO: 20) и Cm-оriC (DCW20) (SEQ ID NO: 50), которые содержали оriC и пару последовательностей ter, встроенных за пределами оriC, соответственно. Последовательность ter представляет собой последовательность, с которой связывается белок Тus. Белок Тus выполняет функцию остановки репликации по механизму, специфичному к направлению последовательности. Термин «встраивание за пределами оriC», если он относится к последовательности ter, означает, что последовательность ter встраивают так, чтобы благодаря действию комбинации белков, которые связываются с последовательностью ter и ингибируют репликацию, репликация осуществлялась по направлению от оriC и доходя до оriC, останавливалась. Cm-оriC (DCW20) представляет собой линейный двухцепочечной фрагмент ДНК в 1298 п.о., а область из 60 оснований, простирающаяся от первого по 60-го основания, представляет собой гомологичную область для связывания с DCW20, которая состоит из такой же последовательности оснований, как и последовательность из 60 оснований от 532-го до 591-го основания DCW20. Область из 60 оснований от 1239-го до 1298-го основания Cm-Oric (DCW20) представляет собой область для связывания с DCW1, которая состоит из такой же последовательности оснований, как и последовательность из 60 оснований от 1-го до 60-го основания DCW1. То есть, если все фрагменты DCW1 - DCW20 (21 фрагмент) и Cm-Oric (DCW20) были связанными, то была получена кольцевая ДНК.
[0142] Кроме того, в качестве связанных линейных двухцепочечных фрагментов ДНК использовали набор из DCW1-DCW25 (SEQ ID NO: 1 - SEQ ID NO: 25) и Cm-оriC (DCW25) (SEQ ID NO: 51), которые включали оriC. Cm-оriC (DCW25) представляет собой линейный двухцепочечной фрагмент ДНК размером 1298 п.о., а область из 60 оснований, простирающаяся от первого по 60-го основания, представляет собой гомологичную область для связывания с DCW25, которая состоит из такой же последовательности оснований, как и последовательность из 60 оснований от 532-го до 591-го основания DCW25. Область из 60 оснований от 1239-го до 1298-го основания Cm-Oric (DCW25) представляет собой область для связывания с DCW1, которая состоит из такой же последовательности оснований, как и последовательность из 60 оснований от 1-го до 60-го основания DCW1. То есть, если все 26 фрагментов DCW1 - DCW25 и Cm-Oric (DCW25) были связанными, то была получена кольцевая ДНК.
[0143] В качестве белка рекомбиназы семейства RecA использовали мутент F203W RecA E. coli, а в качестве 3'→5'-экзонуклеазы использовали экзонуклеазу III. Кроме того, в качестве реакционного раствора для RCR-амплификации использовали смешанный раствор, содержащий 60 нМ Тus в реакционной смеси, состав которой приводится в Таблице 1. Тus получали и очищали из штамма E. coli, экспрессирующего Тus, способом, включающим аффинную колоночную хроматографию и колоночную гель-фильтрацию.
[0144] [Таблица 1]
[0145] В таблице 1 SSB означает SSB, происходящий от E. coli; IHF означает комплекс IhfA и IhfB, происходящий от E. coli; DnaG означает DnaG, происходящий от E. coli; DnaN означает DnaN, происходящий от E. coli; PolIII* означает комплекс ДНК-полимеразы III*, состоящий из DnaX, HolA, HolB, HolC, HolD, DnaE, DnaQ и HolE, происходящих от E. coli; DnaB означает DnaB, происходящий от E. coli; DnaC означает DnaC, происходящий от E. coli; DnaA означает DnaA, происходящий от E. coli; РНКаза H означает РНКазу H, происходящую от E. coli; лигаза представляет собой ДНК-лигазу, происходящую от E. coli; PolI означает ДНК-полимеразу I, происходящую от E. coli; GyrA означает GyrA происходящую от E. coli; GyrB означает GyrB, происходящую от E. coli; Topo IV означает комплекс ParC и ParE, происходящий от E. coli; Topo III означает топоизомеразу III, происходящую от E. coli; а RecQ означает RecQ, происходящую от E. coli.
[0146] SSB был очищен и получен из штамма E. coli, экспрессирующего SSB, способом, включающим преципитацию сульфатом аммония и ионообменную колоночную хроматографию.
IHF получали из штаммов E. coli, коэкспрессирующих IhfA и IhfB, путем очистки, включая преципитацию сульфатом аммония и аффинную колоночную хроматографию.
DnaG получали путем очистки из штамма E. coli, экспрессирующего DnaG, в стадиях, которые включают преципитацию сульфатом аммония, анионообменную колоночную хроматографию и колоночную хроматографию методом гель-фильтрации.
DnaN очищали и получали из штамма E. coli, экспрессирующего DnaN, способом, включающим преципитацию сульфатом аммония и анионообменную колоночную хроматографию.
PolIII* получали и очищали из штаммов E. coli, коэкспрессирующих DnaX, HolA, HolB, HolC, HolD, DnaE, DnaQ и HolE, в стадиях, включающих преципитацию сульфатом аммония, аффинную колоночную хроматографию и колоночную хроматографию методом гель-фильтрации.
DnaB и DnaC получали и очищали из штаммов E. coli, коэкспрессирующих DnaB и DnaC, в стадиях, включающих преципитацию сульфатом аммония, аффинную колоночную хроматографию и колоночную хроматографию методом гель-фильтрации.
DnaA получали и очищали из штамма Escherichia coli, экспрессирующего DnaA, в стадиях, включающих преципитацию сульфатом аммония, преципитацию путем диализа и колоночную хроматографию методом гель-фильтрации.
GyrA и GyrB очищали и получали из смеси штамма E. coli, экспрессирующего GyrА, и штамма E. coli, экспрессирующего GyrB, способом, включающим преципитацию сульфатом аммония, аффинную колоночную хроматографию и колоночную хроматографию методом гель-фильтрации.
Topo IV получали из смеси штамма Escherichia coli, экспрессирующего ParC, и штамма Escherichia coli, экспрессирующего ParE, способом, включающим преципитацию сульфатом аммония, аффинную колоночную хроматографию и колоночную хроматографию методом гель-фильтрации.
Topo III получали из штамма E. coli, экспрессирующего Topo III, путем очистки способом, включающим преципитацию сульфатом аммония и аффинную колоночную хроматографию.
RecQ получали путем очистки из штамма Escherichia coli, экспрессирующего RecQ, в стадиях, включающих преципитацию сульфатом аммония, аффинную колоночную хроматографию и колоночную хроматографию методом гель-фильтрации.
РНКаза H, лигаза и PolI были использованы в виде коммерчески доступных ферментов, происходящих от E. coli (изготавливаемых Takara Bio Inc.).
[0147] В частности, сначала приготавливали реакционные растворы, состоящие из 2,5 нМ или 5 нМ набора линейных двухцепочечных фрагментов ДНК, 1 мкМ мутанта F203W RecA, 80 мЕд/мкл экзонуклеазы III, 20 мМ Триса-HCl (рН 8,0), 4 мМ DТТ, 1 мМ ацетата магния, 100 мкМ АТФ, 4 мМ фосфата креатина, 20 нг/мкл креатинкиназы, 50 мМ глутамата калия, 5% по массе ПЭГ8000 и 10% по объему ДМСО. В качестве набора линейных двухцепочечных фрагментов ДНК использовали набор, содержащий все эквимолярные количества DCW1 - DCW20 и Cm-оriС (DCW20), или набор, содержащий все эквимолярные количества DCW1 - DCW25 и Cm-оriС (DCW25). Затем, эти реакционные растворы инкубировали при 42°C в течение 30 минут для проведения реакции связывания, а затем инкубировали при 65°C в течение 20 минут для термообработки, после чего быстро охлаждали на льду. После термообработки и быстрого охлаждения, 1 мкл реакционных растворов, содержащих 2,5 нМ набора линейных двухцепочечных фрагментов ДНК и 0,5 мкл реакционных растворов, содержащих 5 нМ набора линейных двухцепочечных фрагментов ДНК, подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0148] Результаты окрашивания представлены на фиг. 12(а). На этой фигуре, «1-20» означает дорожку, где использовали раствор, содержащий набор линейных двухцепочечных фрагментов ДНК со всеми эквимолярными количествами DCW1 - DCW20 и Cm-оriC (DCW20). На этой фигуре, «1-25» означает дорожку, где использовали раствор, содержащий набор линейных двухцепочечных фрагментов ДНК со всеми эквимолярными количествами DCW1 - DCW25 и Cm-оriC (DCW25). В результате было подтверждено, что во всех реакционных растворах содержатся связанные молекулы всех размеров.
[0149] Затем, реакционные смеси получали путем добавления, после термообработки и быстрого охлаждения, 0,5 мкл реакционного раствора к 4,5 мкл раствора для реакции RCR-амплификации. Реакционные смеси инкубировали при 30°C в течение 13 часов для проведения RCR-амплификации. После завершения реакции, 1 мкл реакционной смеси подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0150] Результаты окрашивания представлены на фиг. 12(b). На этой фигуре, «1-20» и «1-25» означают такие же дорожки, как и на фиг 12(а). В результате, на дорожке продукта RCR-амплификации в реакционном растворе, в котором присутствовал набор связанных DCW1 - DCW20 и Cm-оriC (DCW20), была детектирована полоса суперспирализованной формы кольцевой связанной молекулы из 21 фрагмента (обозначенной на этой фигуре как «суперспираль» из 21 фрагмента). На дорожке продукта RCR-амплификации в реакционном растворе, в котором присутствовал набор связанных DCW1 - DCW25 и Cm-оriC (DCW25), была детектирована полоса суперспирализованной формы кольцевой связанной молекулы из 26 фрагментов (обозначенной на этой фигуре как «суперспираль» из 26 фрагментов). Хотя после реакции связывания, в реакционном растворе детектировалось много полос (фиг. 12 (а)), однако, после RCR-амплификации, в реакционной смеси детектировалось лишь несколько полос (фиг. 12 (b)). Было подтверждено, что в результате RCR-амплификации были амплифицированы только кольцевые связанные молекулы.
[0151] [Пример 11]
Линейные двухцепочечные фрагменты ДНК двух или более типов были связаны с использованием белка рекомбиназы семейства RecA и 3'→5'-экзонуклеазы с образованием связанной кольцевой молекулы, и полученная связанная кольцевая молекула была подвергнута RCR-амплификации. В этой реакции оценивали влияние термообработки и быстрого охлаждения до RCR амплификации.
[0152] В качестве связанных линейных двухцепочечных фрагментов ДНК использовали набор линейных двухцепочечных фрагментов ДНК со всеми эквимолярными количествами DCW1 - DCW25 и Cm-оriC (DCW25)(называемых здесь «набором из 20 нМ линейных двухцепочечных фрагментов ДНК») Примера 10. В качестве белка рекомбиназы семейства RecA использовали мутант F203W RecA E. coli, а в качестве 3'→5'-экзонуклеазы использовали экзонуклеазу III. Кроме того, в качестве реакционного раствора для RCR-амплификации использовали смешанный раствор, содержащий 60 нМ Тus в реакционной смеси, состав которой приводится в Таблице 1.
[0153] В частности, сначала приготавливали реакционные растворы, состоящие из 20 нМ набора линейных двухцепочечных фрагментов ДНК, 1,5 мкМ мутанта F203W RecA, 80 мЕд/мкл экзонуклеазы III, 20 мМ Триса-HCl (рН 8,0), 4 мМ DТТ, 1 мМ ацетата магния, 100 мкМ АТФ, 4 мМ фосфата креатина, 20 нг/мкл креатинкиназы, 50 мМ глутамата калия, 150 мМ ТМАС, 5% по массе ПЭГ8000 и 10% по объему ДМСО. Затем, эти реакционные растворы инкубировали при 42°C в течение 30 минут для проведения реакции связывания, а затем инкубировали при 50°C или 65°C в течение 2 минут для термообработки, после чего быстро охлаждали на льду. После быстрого охлаждения, 1,5 мкл реакционных растворов подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0154] Результаты окрашивания представлены на фиг. 13(а). На этой фигуре, «500 п.о.-лэддер» означает дорожку, где был использован маркер ДНК-лэддера в Примере 1, а «Вход» означает дорожку, где используется 1,5 мкл раствора, содержащего 20 нМ набора линейных двухцепочечных фрагментов ДНК. «-» означает дорожку, где реакционный раствор не подвергался термообработке после реакции связывания. «50°С» соответствует дорожке, где реакционный раствор был подвергнут термообработке при 50°С в течение 2 минут. «65°С» соответствует дорожке, где реакционный раствор был подвергнут термообработке при 65°С в течение 2 минут. Как показано на фиг. 13 (а), в реакционных растворах без термообработки, ДНК не мигрировала с образованием размытой полосы, тогда как в реакционных растворах, подвергнутых термообработке, большинство размытых полос было удалено.
[0155] Затем, реакционные смеси получали путем добавления, после термообработки и быстрого охлаждения, 0,5 мкл реакционного раствора к 4,5 мкл раствора для реакции RCR-амплификации. Реакционные смеси инкубировали при 30°C в течение 13 часов для проведения RCR-амплификации. В качестве контроля, 0,5 мкл реакционного раствора, после термообработки при 65°С и быстрого охлаждения, добавляли к 4,5 мкл раствора ТЕ, состоящего из 10 мМ Трис-HCl (рН 8,0) и 1 мМ EDTA, с получением раствора для предварительной амплификации. После завершения реакции, 1 мкл раствора для предварительной амплификации и реакционной смеси подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0156] Результаты окрашивания представлены на фиг. 13 (b). На этой фигуре, «MK3» означает дорожку, где использовали маркер ДНК-лэддера. В результате, в реакционной смеси, полученной посредством RCR-амплификации реакционного раствора, где кольцевые связанные молекулы образовывались в результате реакции связывания, была детектирована полоса суперспирализованной формы кольцевой связанной молекулы из 26 фрагментов (обозначенной на этой фигуре как «ссДНК из 25 фрагментов»)(«-», «50°С», «65°С» на фиг. 13(b)). В растворе для предварительной амплификации, полоса не детектировалась («Вход» на фиг. 13(b)). Две широких полосы, где расстояние миграции было больше, чем для полосы суперспирализованной связанной молекулы из 26 фрагментов, детектировались в реакционной смеси без термообработки («-»), тогда как в реакционной смеси, подвергнутой термообработке при 50°С («50°С»), эти полосы были тонкими, и вообще не детектировались при термообработке при 65°С («65°С»). Исходя из этих результатов было обнаружено, что полосы ДНК, где расстояние миграции было больше, чем для полосы суперспирализованной связанной молекулы из 26 фрагментов, соответствовали продуктам амплификации кольцевых связанных молекул, полученных путем неспецифического связывания, и такие продукты неспецифической амплификации могут ингибироваться путем термообработки и быстрого охлаждения до RCR-амплификации.
[0157] [Пример 12]
Линейные двухцепочечные фрагменты ДНК 26 или 36 типов были связаны с использованием белка рекомбиназы семейства RecA и 3'→5'-экзонуклеазы с образованием связанной кольцевой молекулы, и полученная связанная кольцевая молекула была подвергнута RCR-амплификации.
[0158] В качестве связанных линейных двухцепочечных фрагментов ДНК использовали набор из DCW1-DCW25 (SEQ ID NO: 1 - SEQ ID NO: 25) и Km-оriC (DCW25) (SEQ ID NO: 52), которые включали оriC. Km-оriC (DCW25) представляет собой линейный двухцепочечной фрагмент ДНК размером 1509 п.о., а область из 60 оснований, простирающаяся от первого до 60-го основания, представляет собой гомологичную область для связывания с DCW25, которая состоит из такой же последовательности оснований, как и последовательность из 60 оснований от 532-го до 591-го основания DCW25. Область из 60 оснований от 1450-го до 1509-го основания Km-оriC (DCW25) представляет собой область для связывания с DCW1, которая состоит из такой же последовательности оснований как и последовательность из 60 оснований от 1-го до 60-го основания DCW1. То есть, кольцевая ДНК была получена путем связывания всех 26 типов фрагментов DCW1 - DCW25 и Km-оriC (DCW25).
[0159] В качестве связанных линейных двухцепочечных фрагментов ДНК использовали набор из DCW1-DCW35 (SEQ ID NO: 1 - SEQ ID NO: 35) и Km-оriC (DCW35) (SEQ ID NO: 53), которые включали оriC и пару последовательностей ter, встроенных за пределами оriC, соответственно. Km-оriC (DCW35) представляет собой линейный двухцепочечной фрагмент ДНК размером 1509 п.о., а область из 60 оснований, простирающаяся от первого до 60-го основания, представляет собой гомологичную область для связывания с DCW35, которая состоит из такой же последовательности оснований как и последовательность из 60 оснований от 532-го до 591-го основания DCW35. Область из 60 оснований от 1450-го до 1509-го основания Km-оriC (DCW35) представляет собой область для связывания с DCW1, которая состоит из такой же последовательности оснований, как и последовательность из 60 оснований от 1-го до 60-го основания DCW1. То есть, кольцевая ДНК была получена путем связывания всех 36 типов фрагментов DCW1 - DCW35 и Km-оriC (DCW35).
[0160] В качестве белка рекомбиназы семейства RecA использовали мутант F203W RecA E. coli, а в качестве 3'→5'-экзонуклеазы использовали экзонуклеазу III. Кроме того, в качестве реакционного раствора для RCR-амплификации использовали смешанный раствор, содержащий 60 нМ Тus в реакционной смеси, состав которой приводится в Таблице 1.
[0161] В частности, сначала приготавливали реакционные растворы, состоящие из 20 нМ набора линейных двухцепочечных фрагментов ДНК, 1,5 мкМ мутанта F203W RecA, 80 мЕд/мкл экзонуклеазы III, 20 мМ Триса-HCl (рН 8,0), 4 мМ DТТ, 1 мМ ацетата магния, 100 мкМ АТФ, 4 мМ фосфата креатина, 20 нг/мкл креатинкиназы, 50 мМ глутамата калия, 150 мМ ТМАС, 5% по массе ПЭГ8000 и 10% по объему ДМСО. В качестве набора линейных двухцепочечных фрагментов ДНК использовали набор, содержащий все эквимолярные количества DCW1 - DCW25 и Cm-оriC (DCW25) или набор, содержащий все эквимолярные количества DCW1 - DCW35 и Cm-оriC (DCW35). Затем, эти реакционные растворы инкубировали при 42°C в течение 30 минут для проведения реакции связывания, а затем инкубировали при 65°C в течение 5 минут для термообработки, после чего быстро охлаждали на льду. После термообработки и быстрого охлаждения, 1,5 мкл реакционных растворов подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0162] Результаты окрашивания представлены на фиг. 14(а). На этой фигуре, «Вход» означает дорожку, где используется 1,5 мкл раствора, содержащего набор из 20 нМ линейных двухцепочечных фрагментов ДНК. «DCW1-25 Km-оriC» означает дорожку, где используется реакционный раствор, содержащий набор со всеми эквимолярными количествами DCW1 - DCW25 и Km-оriC (DCW25). «DCW1-35 Km-оriC» означает дорожку, где используется реакционный раствор, содержащий набор со всеми эквимолярными количествами DCW1 - DCW35 и Km-оriC (DCW35). Как показано на фиг. 14(а), при использовании любого набора линейных двухцепочечных фрагментов ДНК, связанные молекулы из множества фрагментов были получены посредством реакции связывания.
[0163] Затем 0,5 мкл реакционного раствора, после термообработки и быстрого охлаждения, добавляли к 4,5 мкл раствора для реакции RCR-амплификации с получением реакционной смеси. Реакцию RCR-амплификации осуществляли путем инкубирования реакционной смеси при 30°C в течение 16 часов. Затем 0,5 мкл каждого продукта реакции RCR-амплификации разводили до 4,5 мкл реакционным буфером, который получали путем удаления только группы ферментов из реакционной смеси, представленной в Таблице 1, а затем снова инкубировали при 30°C в течение 30 минут. Обработка путем повторного инкубирования после разведения стимулирует реакцию удлинения репликации и разделения промежуточного продукта амплификации в указанном продукте и способствует повышению количества продуцируемой суперспирализованной ДНК, которая является конечным продуктом. 0,5 мкл реакционного раствора, в которой осуществляют реакцию связывания с использованием DCW1 - DCW25 с последующей термообработкой и быстрым охлаждением, добавляли к 4,5 мкл раствора ТЕ, состоящего из 10 мМ Трис-HCl (рН 8,0) и 1 мМ EDTA. Таким образом, этот полученный раствор использовали в качестве контроля. Этот полученный раствор был использован в качестве контроля в целях получения раствора для предварительной амплификации. После повторного инкубирования, 2,5 мкл раствора для предварительной амплификации и реакционной смеси подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0164] Результаты окрашивания представлены на фиг. 14(b). В результате, в реакционной смеси, полученной посредством RCR-амплификации после связывания набора линейных двухцепочечных фрагментов ДНК, содержащего 26 фрагментов, была детектирована полоса суперспирализованной формы кольцевой связанной молекулы из 26 фрагментов (обозначенной на этой фигуре как «ссДНК из 26 фрагментов») («DCW1-25 Km-оriC» на фиг. 13 (b)). В реакционной смеси, полученной посредством RCR-амплификации после связывания набора линейных двухцепочечных фрагментов ДНК, содержащего 36 фрагментов, была детектирована полоса суперспирализованной формы кольцевой связанной молекулы из 36 фрагментов (обозначенной на этой фигуре как «ссДНК из 36 фрагментов») («DCW1-35 Km-оriC» на фиг. 13 (b)). В растворе для предварительной амплификации, полоса не детектировалась («Вход» на фиг. 14(b)). Исходя из этих результатов было подтверждено, что кольцевая связанная молекула из множества фрагментов, а именно, из 36 фрагментов, может быть получена способом согласно изобретению, и эта кольцевая связанная молекула может быть амплифицирована посредством RCR-амплификации. Однако, связанная молекула из 36 фрагментов, в отличие от связанной молекулы из 26 фрагментов, имела большее число продуктов неспецифической амплификации, полученных посредством RCR-амплификации.
[0165] [Пример 13]
Набор «NEBuilder HiFi для сборки ДНК» (изготавливаемый NEB), используемый в способе связывания множества двухцепочечных фрагментов ДНК методом сборки Гибсона (патентный документ 3), является коммерчески доступным. В этом наборе, линейные двухцепочечные фрагменты ДНК двух или более типов, имеющие гомологичную область из 15-20 оснований на конце, связывали методом NEB, то есть, путем добавления фрагментов ДНК в смешанный раствор (Master Mix) с последующим инкубированием смешанного раствора при 50°С в течение 15-60 минут. Смешанный раствор поставлялся вместе с набором и содержал 5'→3'-экзонуклеазу, ДНК-полимеразу и ДНК-лигазу.
[0166] Эффективность связывания в способе получения ДНК согласно изобретению, где реакцию связывания осуществляли с использованием белка рекомбиназы семейства RecA и 3'→5'-экзонуклеазы, была сравнимой с эффективностью в методе NEB.
[0167] В качестве связанных линейных двухцепочечных фрагментов ДНК использовали набор, содержащий все эквимолярные количества DCW1 - DCW25 (SEQ ID NО:1-SEQ ID NО:25) и Km-оriC (DCW25), который был использован в Примере 12. В качестве белка рекомбиназы семейства RecA использовали мутант F203W RecA E. coli, а в качестве 3'→5'-экзонуклеазы использовали экзонуклеазу III. Кроме того, в качестве реакционного раствора для RCR-амплификации использовали смешанный раствор, содержащий 60 нМ Тus в реакционной смеси, состав которой приводится в Таблице 1.
[0168] В частности, сначала приготавливали реакционные растворы, состоящие из 20 нМ или 60 нМ набора линейных двухцепочечных фрагментов ДНК, 1,5 мкМ мутанта F203W RecA, 80 мЕд/мкл экзонуклеазы III, 20 мМ Триса-HCl (рН 8,0), 4 мМ DТТ, 1 мМ ацетата магния, 100 мкМ АТФ, 4 мМ фосфата креатина, 20 нг/мкл креатинкиназы, 50 мМ глутамата калия, 150 мМ ТМАС, 5% по массе ПЭГ8000 и 10% по объему ДМСО, для их применения в способе согласно изобретению (в способе RA). Затем, эти реакционные растворы инкубировали при 42°C в течение 30 минут для проведения реакции связывания, а затем инкубировали при 65°C в течение 5 минут для термообработки, после чего быстро охлаждали на льду. После быстрого охлаждения, 1,5 мкл реакционных растворов подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0169] В способе NEB, реакционный раствор получали путем смешивания 20 нМ или 60 нМ набора линейных двухцепочечных фрагментов ДНК с раствором, 2-кратно разведенным «смесью Master Mix», поставляемой вместе с указанным набором. Затем, реакционный раствор инкубировали при 50°C в течение 60 минут для проведения реакции связывания. После реакции связывания, 1,5 мкл реакционного раствора подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0170] Результаты окрашивания представлены на фиг. 15(а). На этой фигуре, «Вход» означает дорожку, где используется 1,5 мкл раствора, содержащего набор из 20 нМ линейных двухцепочечных фрагментов ДНК. «RА» означает дорожку, где используется реакционный раствор, полученный способом согласно изобретению (способом RА). «NEB» означает дорожку, где используется реакционный раствор, полученный способом NEB. Как показано на фиг. 15(а), при проведении реакции связывания способом RА было получено значительное число фрагментов независимо от того, имеет ли набор линейных двухцепочечных фрагментов ДНК в реакционном растворе концентрацию 20 нМ или 60 нМ. С другой стороны, в реакционном растворе, где реакцию связывания осуществляют способом NEB, были получены только связанные молекулы из 2-3 фрагментов.
[0171] Затем, реакционные смеси получали, после термообработки и быстрого охлаждения, путем добавления 0,5 мкл реакционного раствора к 4,5 мкл раствора для реакции RCR-амплификации. Реакционные смеси инкубировали при 30°C в течение 16 часов для проведения RCR-амплификации. Затем, 0,5 мкл каждого продукта реакции RCR-амплификации разводили до 4,5 мкл реакционным буфером, который получали путем удаления только группы ферментов из реакционной смеси, представленной в Таблице 1, а затем снова инкубировали при 30°C в течение 30 минут. После повторного инкубирования 2,5 мкл реакционной смеси подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0172] Результаты окрашивания представлены на фиг. 15(b). В результате, в реакционной смеси, полученной посредством RCR-амплификации после реакции связывания способом согласно изобретению (способом RА)(на этой фигуре «RА»), была детектирована полоса суперспирализованной формы кольцевой связанной молекулы, где все 26 фрагментов были связанными (на этой фигуре, «суперспираль из 25 фрагментов»). С другой стороны, в реакционной смеси, полученной посредством RCR-амплификации после реакции связывания способом NEB)(на этой фигуре «NEB»), полоса кольцевой связанной молекулы из 26 фрагментов не детектировалась, и 26 фрагментов не могли быть связаны способом NEB. В обеих реакционных смесях, продукты, которые становятся конкатемерами из-за прохождения неспецифической репликации по типу «катящегося кольца», и кольцевые продукты ДНК-мультимеров (катенаны), которые остаются неразделенными после репликации, также детектировалась с ограничением разделения посредством электрофореза в агарозном геле (на этой фигуре, «мультимер»).
[0173] [Пример 14]
Длинные геномные фрагменты были связаны друг с другом с образованием кольцевой связанной молекулы, и кольцевую связанную молекулу подвергали RCR-амплификации.
[0174] В качестве длинноцепочечного геномного фрагмента использовали Xba I-гидролизат (15 фрагментов, DGF-298/XbaI) геномной ДНК штамма E. coli (DGF-298WΔ100::revΔ234::SC). Из этих гидролизатов, геномный фрагмент 325 т.п.о. (325k-геномный фрагмент) и геномный фрагмент 220 т.п.о. (220k-геномный фрагмент) были связаны друг с другом посредством связывающего фрагмента, содержащего oriC (Cm-оriC-фрагмент) с получением продукта в кольцевой форме. В качестве связывающего фрагмента для циклизации 325k-геномного фрагмента использовали 1298 п.о. линейного двухцепочечного фрагмента ДНК, включающего оriC (Cm-оriC/325k-фрагмента, SEQ ID NО:59). Вышерасположенная концевая область этого связывающего фрагмента была гомологична нижерасположенной концевой области 325k-геномного фрагмента (т.е. 60 оснований, находящихся в вышерасположенном конце этого фрагмента, состояли из такой же последовательности оснований, как и 60 оснований в нижерасположенном конце), а нижерасположенная концевая область этого связывающего фрагмента была гомологична вышерасположенному концу 325k-геномного фрагмента (т.е. 60 оснований, находящихся в нижерасположенном конце этого фрагмента, состояли из такой же последовательности оснований, как и 60 оснований в вышерасположенном конце). В качестве связывающего фрагмента для циклизации 220k-геномного фрагмента использовали 1298 п.о. линейного двухцепочечного фрагмента ДНК, включающего оriC (Cm-оriC/220k-фрагмента, SEQ ID NО:60). Вышерасположенная концевая область этого связывающего фрагмента была гомологична нижерасположенной концевой области 220k-геномного фрагмента (т.е., 60 оснований, находящихся в вышерасположенном конце этого фрагмента, состояли из такой же последовательности оснований, как и 60 оснований в нижерасположенном конце), а нижерасположенная концевая область этого связывающего фрагмента была гомологична вышерасположенному концу 220k-геномного фрагмента (т.е., 60 оснований, находящихся в нижерасположенном конце этого фрагмента, состояли из такой же последовательности оснований, как и 60 оснований в вышерасположенном конце). Кроме того, реакционная смесь, состав которой указан в Таблице 1, была использована в качестве раствора для реакции RCR-амплификации.
[0175] В частности, Xba I-гидролизат геномной ДНК E. coli (DGF-298/XbaI, 4,8 нг/мкл) и Cm-оriC/325k-фрагмент, имеющий область, гомологичную области 325k-геномного фрагмента, который представляет собой геномный фрагмент-мишень (240 пМ), добавляли к реакционной смеси RA [20 мМ Трис-HCl (рН 8,0), 4 мМ DТТ, 150 мМ КОАс, 10 мМ Mg(OAc)2, 100 мкМ АТФ, 5% по массе ПЭГ8000, 40 мЕд/мкл экзонуклеазы III, 1 мкМ мутанта F203W RecA E. coli] (5 мкл), и раствор инкубировали при 30°C в течение 60 минут для проведения реакции связывания. 0,5 мкл полученного продукта RA добавляли к раствору для реакции RCR-амплификации (4,5 мкл), и реакцию амплификации осуществляли с использованием термоцикла (1 цикл при 37°C в течение 1 минуты, а затем при 24°C в течение 30 минут, всего 40 циклов). Реакцию связывания проводили аналогичным способом, но с использованием Cm-оriC/220k-фрагмента вместо Cm-оriC/325k-фрагмента, и включением геномного фрагмента-мишени размером 220 т.п.о., а затем проводили реакцию RCR-амплификации. В качестве контроля, кольцевую плазмиду оriC размером 200 т.п.о. подвергали RCR-амплификации. К раствору реакции RCR-амплификации продукта связывания 325k-геномного фрагмента добавляли 50 мкМ диэтилентриаминпентауксусной кислоты для стабилизации длинноцепочечной ДНК.
[0176] После реакции, 1 мкл реакционной смеси подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR. Результаты окрашивания представлены на фиг. 16. На этой фигуре «220 т.п.о.» означает дорожку, где используется продукт амплификации, полученный посредством реакции с геномным фрагментом-мишенью размером 220 т.п.о. «325 т.п.о.» означает дорожку, где используется продукт амплификации, полученный посредством реакции с геномным фрагментом-мишенью размером 325 т.п.о. «200 т.п.о. (RCR-контроль)» означает дорожку, где используется продукт, полученный путем прямой амплификации кольцевой плазмиды оriC размером 200 т.п.о. В результате, суперспирализованная форма кольцевой связанной молекулы размером 220 т.п.о. и суперспирализованная форма кольцевой связанной молекулы размером 325 т.п.о. были детектированы в результате реакции с геномным фрагментом-мишенью размером 220 т.п.о. и в результате реакции с геномным фрагментом-мишенью размером 325 т.п.о., соответственно. Исходя из этих результатов было обнаружено, что двухцепочечный фрагмент ДНК размером по меньшей мере 325 т.п.о. может быть циклизован способом получения ДНК согласно изобретению.
[0177] [Пример 15]
Линейные двухцепочечные фрагменты ДНК двух или более типов были связаны с использованием белка рекомбиназы семейства RecA и 3'→5'-экзонуклеазы. В этой реакции было исследовано влияние концентрации фосфата креатина в реакционном растворе, содержащем АТФ-регенерирующую систему, состоящую из креатинкиназы и фосфата креатина.
[0178] DCW1-DCW10 (SEQ ID NO: 1 - SEQ ID NO: 10) были использованы в качестве связанных линейных двухцепочечных фрагментов ДНК. В качестве белка рекомбиназы семейства RecA использовали мутант F203W RecA E. coli, а в качестве 3'→5'-экзонуклеазы использовали экзонуклеазу III.
[0179] В частности, сначала приготавливали реакционные растворы, состоящие из 2 нМ каждого из DCW1-DCW10, 1,5 мкМ мутанта F203W RecA, 80 мЕд/мкл экзонуклеазы III, 20 мМ Триса-HCl (рН 8,0), 4 мМ DТТ, 1 мМ ацетата магния, 50 мМ глутамата калия, 100 мкМ АТФ, 150 мМ ТМАС, 5% по массе ПЭГ8000, 10% по объему ДМСО, 20 нг/мкл креатинкиназы и 0 мМ (без добавления), 0,1 мМ, 0,4 мМ, 1 мМ, 4 мМ или 10 мМ фосфата креатина. Затем эти реакционные растворы инкубировали при 42°C в течение 30 минут для проведения реакции связывания, а затем инкубировали при 65°C в течение 2 минут для термообработки, после чего быстро охлаждали на льду. После термообработки и быстрого охлаждения, 1,5 мкл реакционных растворов подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0180] Результаты окрашивания представлены на фиг. 17. На этой фигуре «Вход» означает дорожку, где используется 2 мкл раствора, содержащего 2 нМ каждого из DCW1-DCW10. В результате, для образцов, подвергнутых реакции связывания, была детектирована полоса связанной молекулы, полученной путем связывания фрагментов всех 10 типов в образцах с 0,4-10 мМ фосфата креатина. В частности, было обнаружено, что образцы, содержащие 1 мМ или 4 мМ фосфата креатина, имели большое количество связанных молекул из 10 фрагментов, и среди этих образцов, образец, содержащий 4 мМ фосфата креатина, имел большое количество связанных молекул из 2-9 фрагментов, и обнаруживал превосходную эффективность связывания.
[0181] [Пример 16]
Линейные двухцепочечные фрагменты ДНК двух или более типов были связаны с использованием белка рекомбиназы семейства RecA и 3'→5'-экзонуклеазы. В этой реакции было исследовано влияние концентрации креатинкиназы в реакционном растворе, содержащем АТФ-регенерирующую систему, состоящую из креатинкиназы и фосфата креатина.
[0182] DCW1-DCW10 (SEQ ID NO: 1 - SEQ ID NO: 10) были использованы в качестве связанных линейных двухцепочечных фрагментов ДНК. В качестве белка рекомбиназы семейства RecA использовали мутант F203W RecA E. coli, а в качестве 3'→5'-экзонуклеазы использовали экзонуклеазу III.
[0183] В частности, сначала приготавливали реакционные растворы, состоящие из 2 нМ каждого из DCW1-DCW10, 1,5 мкМ мутанта F203W RecA, 80 мЕд/мкл экзонуклеазы III, 20 мМ Триса-HCl (рН 8,0), 4 мМ DТТ, 1 мМ ацетата магния, 50 мМ глутамата калия, 100 мкМ АТФ, 150 мМ ТМАС, 5% по массе ПЭГ8000, 10% по объему ДМСО, 4 мМ фосфата креатина и 0 нг/мкл, 20 нг/мкл, 50 нг/мкл или 200 нг/мкл креатинкиназы. Затем эти реакционные растворы инкубировали при 42°C в течение 30 минут для проведения реакции связывания, а затем инкубировали при 65°C в течение 2 минут для термообработки, после чего быстро охлаждали на льду. После термообработки и быстрого охлаждения, 1,5 мкл реакционных растворов подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0184] Результаты окрашивания представлены на фиг. 18. На этой фигуре, «Вход» означает дорожку, где используется 2 мкл раствора, содержащего 2 нМ каждого из DCW1-DCW10. «Буфер» означает дорожку, где используется образец, в который не добавляли креатинкиназу (0 нг/мкл). В результате, для образцов, подвергнутых реакции связывания, была детектирована полоса связанной молекулы, полученной путем связывания фрагментов всех 10 типов во всех образцах с добавлением креатинкиназы.
[0185] [Пример 17]
Линейные двухцепочечные фрагменты ДНК двух или более типов были связаны с использованием белка рекомбиназы семейства RecA и 3'→5'-экзонуклеазы. В этой реакции было исследовано влияние АТФ-регенерирующей системы, состоящей из пируваткиназы и пирувата фосфоэнола.
[0186] В качестве связанных линейных двухцепочечных фрагментов ДНК использовали набор из DCW1-DCW35 (SEQ ID NO: 1 - SEQ ID NO: 35) и Km-оriC (DCW35) (SEQ ID NO: 53), которые включали оriC и пару последовательностей ter, встроенных за пределами оriC, соответственно. В качестве белка рекомбиназы семейства RecA использовали мутант F203W RecA E. coli, а в качестве 3'→5'-экзонуклеазы использовали экзонуклеазу III.
[0187] В частности, сначала приготавливали реакционные растворы, состоящие из 0,6 нМ каждого из DCW1-DCW35 (SEQ ID NО: - SEQ ID NО: 5) и Km-оriC (DCW35) (SEQ ID NO: 53), 1,5 мкМ мутанта F203W RecA, 80 мЕд/мкл экзонуклеазы III, 20 мМ Триса-HCl (рН 8,0), 4 мМ DТТ, 1 мМ ацетата магния, 50 мМ глутамата калия, 100 мкМ АТФ, 150 мМ ТМАС, 5% по массе ПЭГ8000, 10% по объему ДМСО, 2 мМ пирувата фосфоэнола и 10 нг/мкл, 32 нг/мкл или 100 нг/мкл пируваткиназы. Реакционный раствор для сравнения получали аналогичным образом, за исключением того, что вместо 2 мМ пирувата фосфоэнола использовали 2 мМ фосфата креатина, а вместо пируваткиназы использовали 20 нг/мкл креатинкиназы в виде смеси. Реакционный раствор для сравнения был также получен аналогичным образом, за исключением того, что пируват фосфоэнола и пируваткиназу не использовали. Затем, эти реакционные растворы инкубировали при 42°C в течение 30 минут для проведения реакции связывания, а затем инкубировали при 65°C в течение 2 минут для термообработки, после чего быстро охлаждали на льду. После термообработки и быстрого охлаждения, 1,5 мкл реакционных растворов подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0188] Результаты окрашивания представлены на фиг. 19. На этой фигуре, «Вход» означает дорожку, где используется 2 мкл раствора, содержащего 0,6 нМ каждого из DCW1-DCW35 (SEQ ID NО: 1 - SEQ ID NО: 35) и Km-оriC (DCW35). «АТФ-регенерация» означает дорожку, где используется обрзец без пирувата фосфоэнола и пируваткиназы. «2 мМ CP, 20 нг/мкл CK» означает дорожку, где используется образец, содержащий фосфат креатина и креатинкиназу. «2 мМ PEP» означает дорожку, где используется образец, содержащий пируват фосфоэнола и пируваткиназу. В результате, полосы связанных молекул из множества фрагментов были детектированы в образцах, содержащих АТФ-регенерирующую систему, состоящую из 2 мМ пирувата фосфоэнола и 100 нг/мкл пируваткиназы, по аналогии с образцами, содержащими АТФ-регенерирующую систему, состоящую из фосфата креатина и креатинкиназы.
[0189] [Пример 18]
Линейные двухцепочечные фрагменты ДНК двух или более типов были связаны с использованием белка рекомбиназы семейства RecA и 3'→5'-экзонуклеазы. В этой реакции было исследовано влияние АТФ-регенерирующей системы, состоящей из полифосфаткиназы и полифосфата.
[0182] DCW1-DCW10 (SEQ ID NO: 1 - SEQ ID NO: 10) были использованы в качестве связанных линейных двухцепочечных фрагментов ДНК. В качестве белка рекомбиназы семейства RecA использовали мутант F203W RecA E. coli, а в качестве 3'→5'-экзонуклеазы использовали экзонуклеазу III.
[0183] В частности, сначала приготавливали реакционные растворы, состоящие из 2 нМ каждого из DCW1-DCW10, 1 мкМ RecA дикого типа, 80 мЕд/мкл экзонуклеазы III, 20 мМ Триса-HCl (рН 8,0), 4 мМ DТТ, 1 мМ ацетата магния, 50 мМ глутамата калия, 100 мкМ АТФ, 150 мМ ТМАС, 5% по массе ПЭГ8000, 10% по объему ДМСО, 1 мМ, 4 мМ или 10 мМ полифосфата и 20 нг/мкл, 60 нг/мкл или 150 нг/мкл полифосфаткиназы. Затем эти реакционные растворы инкубировали при 42°C в течение 30 минут для проведения реакции связывания, а затем инкубировали при 65°C в течение 2 минут для термообработки, после чего быстро охлаждали на льду. После термообработки и быстрого охлаждения, 1,5 мкл реакционных растворов подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0184] Результаты окрашивания представлены на фиг. 20. На этой фигуре «Вход» означает дорожку, где используется 2 мкл раствора, содержащего 2 нМ каждого из DCW1-DCW10. В результате, для образцов, подвергнутых реакции связывания, была детектирована полоса связанной молекулы, полученной путем связывания фрагментов всех 10 типов во образце, содержащем 60 нг/мкл полифосфаткиназы и 1 мМ полифосфата.
[0193] [Пример 19]
Линейные двухцепочечные фрагменты ДНК двух или более типов были связаны с использованием белка рекомбиназы семейства RecA и 3'→5'-экзонуклеазы. В этой реакции было исследовано влияние использования комбинации 3'→5'-экзонуклеазы, специфичной к линейной двухцепочечной ДНК, и 3'→5'-экзонуклеазы, специфичной к одноцепочечной ДНК.
[0194] DCW1-DCW10 (SEQ ID NO: 1 - SEQ ID NO: 10) были использованы в качестве связанных линейных двухцепочечных фрагментов ДНК. В качестве 3'→5'-экзонуклеазы, специфичной к линейной двухцепочечной ДНК, и 3'→5'-экзонуклеазы, специфичной к одноцепочечной ДНК, использовали экзонуклеазу III и экзонуклеазу I, соответственно.
[0195] В частности, сначала приготавливали реакционные растворы, состоящие из 1 нМ каждого из DCW1-DCW10, 1 мкМ RecA дикого типа, 80 мЕд/мкл экзонуклеазы III, 20 мМ Триса-HCl (рН 8,0), 4 мМ DТТ, 1 мМ ацетата магния, 50 мМ глутамата калия, 100 мкМ АТФ, 150 мМ ТМАС, 5% по массе ПЭГ8000, 10% по объему ДМСО, 1 мМ, 4 мМ или 10 мМ полифосфата и 20 нг/мкл, 60 нг/мкл или 150 нг/мкл полифосфаткиназы. Затем эти реакционные растворы инкубировали при 42°C в течение 30 минут для проведения реакции связывания, а затем инкубировали при 65°C в течение 2 минут для термообработки, после чего быстро охлаждали на льду. После термообработки и быстрого охлаждения, 1,5 мкл реакционных растворов подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0196] Результаты окрашивания представлены на фиг. 21. На этой фигуре «Вход» означает дорожку, где используется 2 мкл раствора, содержащего 1 нМ каждого из DCW1-DCW10. В результате, во всех образцах, подвергнутых реакции связывания, была детектирована полоса связанной молекулы, полученной путем связывания фрагментов всех 10 типов. Количество связанной молекулы, полученной путем связывания фрагментов всех 10 типов, увеличивалось в зависимости от содержания экзонуклеазы I. Исходя из этих результатов было обнаружено, что добавление экзонуклеазы I стимулирует реакцию связывания с использованием экзонуклеазы III и RecA.
[0197] [Пример 20]
Линейные двухцепочечные фрагменты ДНК 36 типов были связаны с использованием белка рекомбиназы семейства RecA; 3'→5'-экзонуклеазы, специфичной к линейной двухцепочечной ДНК; и 3'→5'-экзонуклеазы, специфичной к одноцепочечной ДНК, с образованием связанной кольцевой молекулы, и эта связанная кольцевая молекула была подвергнута RCR-амплификации.
[0198] В качестве связанных линейных двухцепочечных фрагментов ДНК использовали набор из DCW1-DCW35 (SEQ ID NO: 1 - SEQ ID NO: 35) и Km-оriC (DCW35) (SEQ ID NO: 52), которые включали оriC и пару последовательностей ter, встроенных за пределами оriC, соответственно. В качестве белка рекомбиназы семейства RecA использовали RecA E. coli дикого типа. В качестве 3'→5'-экзонуклеазы специфичной к линейной двухцепочечной ДНК и 3'→5'-экзонуклеазы, специфичной к одноцепочечной ДНК, использовали экзонуклеазу III и экзонуклеазу I, соответственно. Кроме того, в качестве реакционного раствора для RCR-амплификации использовали смешанный раствор, содержащий 60 нМ Тus в реакционной смеси, состав которой приводится в Таблице 1.
[0199] В частности, сначала приготавливали реакционные растворы, состоящие из 0,6 нМ каждого из DCW1-DCW35 и Km-оriC (DCW35), 1,5 мкМ RecA дикого типа, 80 мЕд/мкл экзонуклеазы III, 20 мМ Триса-HCl (рН 8,0), 4 мМ DТТ, 1 мМ ацетата магния, 50 мМ глутамата калия, 100 мкМ АТФ, 150 мМ ТМАС, 5% по массе ПЭГ8000, 10% по объему ДМСО, 20 нг/мкл креатинкиназы, 4 мМ фосфата креатина и 0 ед./мкл (без добавления), 0,3 ед./мкл или 1 ед./мкл экзонуклеазы I. Затем, эти реакционные растворы инкубировали при 42°C в течение 30 минут для проведения реакции связывания, а затем инкубировали при 65°C в течение 2 минут для термообработки, после чего быстро охлаждали на льду. После термообработки и быстрого охлаждения 1,5 мкл реакционных растворов подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0200] Результаты окрашивания представлены на фиг. 22(а). На этой фигуре, «Вход» означает дорожку, где используется 2 мкл раствора, содержащего 0,6 нМ каждого из DCW1-DCW35 и Km-оriC (DCW35). Как показано на фиг. 22(а), связанные молекулы из множества фрагментов детектировались во всех образцах. Образец, в который было добавлено большее количество экзонуклеазы I, имеет большее количество связанных молекул из множества фрагментов.
[0201] Затем, реакционные смеси получали, после термообработки и быстрого охлаждения, путем добавления 0,5 мкл реакционного раствора к 4,5 мкл раствора для реакции RCR-амплификации. Реакционные смеси инкубировали при 30°C в течение 16 часов для проведения RCR-амплификации. Затем, 0,5 мкл каждого продукта реакции RCR-амплификации разводили до 4 мкл реакционным буфером, который получали путем удаления только группы ферментов из реакционной смеси, представленной в Таблице 1, а затем снова инкубировали при 30°C в течение 30 минут. После повторного инкубирования, 2,5 мкл реакционной смеси подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0202] Результаты окрашивания представлены на фиг. 22(b). В результате, в образце, содержащем экзонуклеазу I, была детектирована полоса суперспирализованной формы кольцевой связанной молекулы из 36 фрагментов (обозначенной на этой фигуре как «ссДНК из 36 фрагментов») («суперспираль из 36 фрагментов на фиг. 13 (b)). С другой стороны, в образце без добавления экзонуклеазы I, эта полоса не наблюдалось. Исходя из этих результатов было обнаружено, что эффективность реакции связывания, проводимой с использованием RecA и зкзонуклеазы III, стимулировалась после добавления экзонуклеазы I, что приводило к получению кольцевой связанной молекулы, полученной путем связывания фрагментов всех 36 типов.
[0203] [Пример 21]
Линейные двухцепочечные фрагменты ДНК 50 типов были связаны с использованием белка рекомбиназы семейства RecA; 3'→5'-экзонуклеазы; и 3'→5'-экзонуклеазы, специфичной к одноцепочечной ДНК, с образованием связанной кольцевой молекулы, и эта связанная кольцевая молекула была подвергнута RCR-амплификации.
[0204] В качестве связанных линейных двухцепочечных фрагментов ДНК использовали набор из DCW1-DCW49 (SEQ ID NO: 1 - SEQ ID NO: 49) и Km-оriC (DCW49) (SEQ ID NO: 62), которые включали оriC и пару последовательностей ter, встроенных за пределами оriC, соответственно. Km-оriC (DCW49) представляет собой линейный двухцепочечной фрагмент ДНК размером 1509 п.о., а область из 60 оснований, простирающаяся от первого до 60-го основания, представляет собой гомологичную область для связывания с DCW49, которая состоит из такой же последовательности оснований, как и последовательность из 60 оснований от 532-го до 591-го основания DCW49. Область из 60 оснований от 1450-го до 1509-го основания Km-оriC (DCW49) представляет собой область для связывания с DCW1, которая состоит из такой же последовательности оснований, как и последовательность из 60 оснований от 1-го до 60-го основания DCW1. То есть, кольцевая ДНК была получена путем связывания всех 50 типов фрагментов DCW1 - DCW49 и Km-оriC (DCW49).
[0205] В качестве позитивного контроля использовали набор из DCW1-DCW35 (SEQ ID NO: 1 - SEQ ID NO: 35) и Km-оriC (DCW35), который включал оriC и пару последовательностей ter, встроенных за пределами оriC, соответственно. В качестве белка рекомбиназы семейства RecA использовали RecA E. coli дикого типа. В качестве 3'→5'-экзонуклеазы, специфичной к линейной двухцепочечной ДНК, и 3'→5'-экзонуклеазы, специфичной к одноцепочечной ДНК, использовали экзонуклеазу III и экзонуклеазу I. Кроме того, в качестве реакционного раствора для RCR-амплификации использовали смешанный раствор, содержащий 60 нМ Тus в реакционной смеси, состав которой приводится в Таблице 1.
[0206] В частности, сначала приготавливали реакционные растворы, состоящие из 0,6 нМ каждого из DCW1-DCW49 и Km-оriC (DCW49), 1,5 мкМ RecA дикого типа, 80 мЕд/мкл экзонуклеазы III, 20 мМ Триса-HCl (рН 8,0), 4 мМ DТТ, 1 мМ ацетата магния, 50 мМ глутамата калия, 100 мкМ АТФ, 150 мМ ТМАС, 5% по массе ПЭГ8000, 10% по объему ДМСО, 20 нг/мкл креатинкиназы, 4 мМ фосфата креатина и 0,3 ед./мкл экзонуклеазы I. Реакционный раствор получали аналогичным образом, за исключением того, что вместо 0,6 нМ каждого из DCW1-DCW49 и Km-оriC (DCW49) использовали смесь 0,6 нМ каждого из DCW1-DCW35 и Km-оriC (DCW35). Затем эти реакционные растворы инкубировали при 42°C в течение 30 минут для проведения реакции связывания, а затем инкубировали при 65°C в течение 2 минут для термообработки, после чего быстро охлаждали на льду. После термообработки и быстрого охлаждения, 1,5 мкл реакционных растворов подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0207] Результаты окрашивания представлены на фиг. 23(а). На этой фигуре, для «20 нМ (8,8 нг/мл) DCW1-35 Km-оriC», «Вход» означает дорожку, где используется 1,5 мкл раствора, содержащего 0,6 нМ каждого из DCW1-DCW35 и Km-оriC (DCW35), а «RA» означает дорожку, где используется реакционный раствор, содержащий 0,6 нМ каждого из DCW1-DCW35 и Km-оriC (DCW35). Для «30 нМ (12,1 нг/мл) DCW1-49 Km-оriC», «Вход» означает дорожку, где используется 1,5 мкл раствора, содержащего 0,6 нМ каждого из DCW1-DCW49 и Km-оriC (DCW349, а «RA» означает дорожку, где используется реакционный раствор, содержащий 0,6 нМ каждого из DCW1-DCW49 и Km-оriC (DCW49). В результате, связанные молекулы из множества фрагментов детектировались в образцах, полученных с использованием DCW1-DCW35 и Km-оriC (DCW35), и в образцах, полученных с использованием DCW1-DCW49 и Km-оriC (DCW49).
[0208] Затем реакционные смеси получали, после термообработки и быстрого охлаждения, путем добавления 0,5 мкл реакционного раствора к 4,5 мкл раствора для реакции RCR-амплификации. Реакционные смеси инкубировали при 30°C в течение 16 часов для проведения RCR-амплификации. Затем, 0,5 мкл каждого продукта реакции RCR-амплификации разводили до 4 мкл реакционным буфером, который получали путем удаления только группы ферментов из реакционной смеси, представленной в Таблице 1, а затем снова инкубировали при 30°C в течение 30 минут. После повторного инкубирования, 2,5 мкл реакционной смеси подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0209] Результаты окрашивания представлены на фиг. 23 (b). На этой фигуре, «MK3» означает дорожку, где используется маркер ДНК-лэддера. В результате, как было подтверждено в примере 20, в образце, полученном с использованием DCW1 - DCW35 и Km-оriC (DCW35), была получена кольцевая связанная молекула из 36 фрагментов и был детектирован продукт ее амплификации. С другой стороны, в образцах, полученных с использованием DCW1 - DCW49 и Km-оriC (DCW49), была детектирована тонкая полоса в положении, где ожидается полоса кольцевой связанной молекулы из 50 фрагментов.
[0210] Затем выделяли ДНК, содержащуюся в реакционном растворе, полученном путем реакции RCR-амплификации после реакции связывания с использованием реакционного раствора, содержащего DCW1 - DCW49 и Km-оriC (DCW49) (продукта амплификации связанной кольцевой молекулы, полученной путем связывания 50 фрагментов) и оценивали структуру последовательности оснований ДНК.
[0211] В частности, 9 мкл буфера ТЕ (раствора, содержащего 10 мМ Триса-HCl (рН 8,0) и 1 мМ EDTA) добавляли к 1 мкл раствора после реакции RCR, и 1 мкл полученного разведенного раствора смешивали с 50 мкл раствора, содержащего компетентные клетки E. coli (E. coli HST08 Premium Electro-Cell, от Takara Bio Inc.). Полученную смесь подвергали электропорации и трансформации. Двенадцать колоний полученных трансформантов культивировали в течение ночи в 20 мл жидкой среды LB, содержащей 50 мкг/мл канамицина, и плазмидную ДНК, оставшуюся в клетках Escherichia coli, культивированных в каждом культуральном растворе, экстрагировали. Концентрацию ДНК в полученном экстракте ДНК вычисляли путем измерения оптической плотности на длине волны 260 нм. После вычисления концентрации ДНК, 15 нг экстрагированной ДНК подвергали электрофорезу в агарозном геле и разделенные полосы окрашивали зеленым SYBR.
[0212] В результате, в 3 (№ 6, 8, 10) из 12 колоний детектировалась полоса продукта амплификации связанной молекулы из 50 фрагментов (двухцепочечной кольцевой ДНК без гэпов или ников). Затем приблизительно 15 нг ДНК, экстрагированной из этих 3 колоний и колонии (No. 12), в которой полоса продукта амплификации связанной молекулы из 50 фрагментов не могла быть детектирована, подвергали электрофорезу а агарозном геле с использованием геля, состоящего из 1 масс. % агарозы, и разделенные полосы окрашивали зеленым SYBR. Результаты окрашивания представлены на фиг. 24. На этой фигуре, «MK3» означает дорожку, где используется маркер ДНК-лэддера, а «RCR» означает дорожку, где используется раствор после реакции RCR. «Геном» означает полосу геномной ДНК E. coli, а «*» означает полосу связанной молекулы из 50 фрагментов. Как показано на фиг. 24, в трансформантах, содержащихся в колониях 6, 8 и 10, детектировались только полосы продуктов амплификации геномной ДНК Escherichia coli и связанные молекулы из 50 фрагментов.
[0213] Затем, были оценены структуры последовательности ДНК-мишени, которая, предположительно, представляет собой связанные кольцевые молекулы, полученные путем связывания 50 фрагментов, выделенных их трансформантов колоний № 6, 8 и 10. Исходя из нуклеотидной последовательности связанной молекулы из 50 фрагментов было обнаружено, что гидролиз рестриктирующим ферментом PciI давал всего четыре фрагмента, состоящих из 10849 п.о., 8121 п.о., 4771 п.о. и 3694 п.о., а гидролиз рестриктирующим ферментом NcoI давал всего шесть фрагментов, состоящих из 11308 п.о., 7741 п.о., 4407 п.о., 2599 п.о., 1123 п.о. и 257 п.о.. Таким образом, ДНК-мишень, которая, как предполагается, представляет собой кольцевую связанную молекулу, выделенную из каждого трансформанта, гидролизовали ферментами PciI или NcoI, и оценивали паттерны их полос.
[0214] В частности, 0,5 мкл 0,03 нг/мкл экстрагированной ДНК добавляли к 4,5 мкл раствора реакции RCR-амплификации и получали реакционный раствор. Реакционный раствор инкубировали при 30°C в течение 16 часов для проведения реакции RCR-амплификации. Затем, 5 мкл каждого продукта реакции RCR-амплификации разводили 20 мкл буфера для реакции RCR (в таблице 1, этот буфер указан как «буфер для реакции), после чего снова инкубировали при 30°C в течение 30 минут. После повторного инкубирования, 25 мкл реакционной смеси добавляли к 25 мкл раствора, содержащего 50 мМ Триса-HCl (рН 8,0), 50 мМ EDTA, 0,2 масс.% додецилсульфата натрия, 100 мкг/мл проназы К, 10 масс.% глицерина и 0,2 масс.% бромфенолового синего, а затем инкубировали при 37°C в течение 30 минут для разложения белков реакции RCR. После инкубирования, к раствору добавляли равное количество раствора PCI (ТЕ-насыщенный фенол:хлороформ:изоамиловый спирт = 25:24:1), и смесь интенсивно встряхивали с использованием вихревой мешалки, а затем центрифугировали при 12000 об./мин в течение 1 минуты. Отделенный водный слой диализовали против буфера ТЕ с использованием мембранных фильтров MF (торговый знак)(тип фильтра: 0,05 мкм VMWP, изготовленный Merck). Концентрацию ДНК в растворе ДНК после диализа вычисляли исходя из оптической плотности раствора ДНК на длине волны 260 нм. После диализа получали 4,5 мкл раствора, содержащего 40 нг ДНК, 1 × буфера NEB 3, 0,1 масс.% BSA, а затем к раствору добавляли 0,5 мкл 10 ед./мкл рестриктирующего фермента PciI (изготовитель Takara Bio Inc.), 10 ед./мкл рестриктирующего фермента NcoI (изготовитель New England Biolabs), или воду. Полученный раствор инкубировали при 37°C в течение 30 минут. После инкубирования, 2,5 мкл реакционного раствора подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0215] Результаты окрашивания представлены на фиг. 25. На этой фигуре, «MK3» и «MK2» означают дорожку, где используется маркер ДНК-лэддера, соответственно. «ПЦР-продукт» означает дорожку, где используется раствор после реакции RCR. «6», «8» и «10» означают дорожки, где используются продукты реакции RCR-амплификации ДНК, экстрагированной из трансформантов колоний 6, 8 и 10, соответственно. «-» означает дорожку, где используется образец без обработки ферментом. В результате было подтверждено, что кольцевая ДНК, содержащаяся в трансформантах № 6, 8, 10, представляет собой кольцевые связанные молекулы, в которых 50 представляющих интерес фрагментов были связанными, на что указывали паттерны полос гидролизатов ферментов PciI и NcoI.
[0216] [Пример 22]
Линейные двухцепочечные фрагменты ДНК двух или более типов, содержащие фрагмент ДНК с гомологичной областью, расположенной у выступающего 3'-конца или поблизости от него, были связаны с использованием белка рекомбиназы семейства RecA и 3'→5'-экзонуклеазы. В этой реакции было исследовано влияние комбинации 3'→5'-экзонуклеазы, специфичной к линейной двухцепочечной ДНК, и 3'→5'-экзонуклеазы, специфичной к одноцепочечной ДНК.
[0217] В качестве связанных линейных двухцепочечных фрагментов ДНК использовали линейный двухцепочечный фрагмент ДНК с выступающими 3'-концами, который был получен путем гидролиза pUC4KSceI (фрагмента PUC4KSceI), и линейный двухцепочечный фрагмент ДНК был сконструирован так, чтобы он связывался в форме кольцевой связанной молекулы с этим линейным двухцепочечным фрагментом ДНК (Km-оriC PI-SceI). pUC4KSceI представляет собой плазмиду, полученную путем связывания и циркуляризации 4 т.п.о.-фрагмента и 500 п.о.-PI-SceI-фрагмента (SEQ ID NО:65) с RA. 4 т.п.о.-фрагмент получали путем ПЦР-амплификации с использованием плазмиды PUC4K в качестве матрицы и пары праймеров (CTATGCGGCATCAGAGCAG (SEQ ID NO: 63) и GTTAAGCCAGCCCCGACAC (SEQ ID NO: 64)). Km-оriC PI-SceI представляет собой ПЦР-фрагмент, амплифицированный с использованием фрагмента Km-оriC (DCW35) в качестве матрицы и пары праймеров ((tgcgtaagcggggcacatttcattacctctttctccgcacGCTCTGCCAGTGTTACAACC (SEQ ID NO: 66) и taatgtatactatacgaagttattatctatgtcgggtgc TAACGCGGTATGAAAATGGAT (SEQ ID NO: 67)).
[0218] В качестве белка рекомбиназы семейства RecA использовали мутант F203W RecA E. coli. В качестве 3'→5'-экзонуклеазы, специфичной к линейной двухцепочечной ДНК и 3'→5'-экзонуклеазы, специфичной к одноцепочечной ДНК, использовали экзонуклеазу III и экзонуклеазу I, соответственно.
[0219] В частности, сначала приготавливали 1,28 нМ каждого фрагмента PUC4KSceI и Km-оriC PI-SceI, 1,5 мкМ RecA дикого типа, 80 мЕд/мкл экзонуклеазы III, 20 мМ Триса-HCl (рН 8,0), 4 мМ DТТ, 1 мМ ацетата магния, 50 мМ глутамата калия, 100 мкМ АТФ, 150 мМ ТМАС, 5% по массе ПЭГ8000, 10% по объему ДМСО, 4 мМ фосфата креатина, 20 нг/мкл креатинкиназы и 0 ед./мкл (без добавления), 0,3 ед./мкл, 0,6 ед./мкл или 1 ед./мкл экзонуклеазы I. Затем эти реакционные растворы инкубировали при 42°C в течение 60 минут для проведения реакции связывания, а затем инкубировали при 65°C в течение 5 минут для термообработки, после чего быстро охлаждали на льду. После термообработки и быстрого охлаждения, 1,5 мкл реакционных растворов подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0220] Результаты окрашивания представлены на фиг. 26. На этой фигуре, «Вход» означает дорожку, где используется 2 мкл раствора, содержащего 1,28 нМ каждого фрагмента PUC4KSceI и Km-оriC PI-SceI. На этой фигуре, «рUC4KSceI» означает полосу фрагмента pUC4KSceI, «Km-оriC» означает полосу Km-оriC PI-SceI, а «продукт сборки» означает полосу связанной молекулы, полученной путем связывания фрагмента рUC4KSceI и Km-оriC PI-SceI. В результате, образец, в который добавляли большее количество экзонуклеазы I, имел большее количество связанных молекул, состоящих из фрагментов. Было подтверждено, что эффективность связывания повышалась с использованием экзонуклеазы I, гидролизующей выступающие 3'-концы, в отличие от экзонуклеазы III, которая дает выступающие 5'-концы, которые могут быть легко получены в качестве мишени с использованием экзонуклеазы III.
[0221] [Пример 23]
Линейные двухцепочечные фрагменты ДНК двух или более типов были связаны с использованием белка рекомбиназы семейства RecA и 3'→5'-экзонуклеазы. В этой реакции было исследовано влияние комбинации 3'→5'-экзонуклеазы, специфичной к линейной двухцепочечной ДНК, и двух типов 3'→5'-экзонуклеаз, специфичных к одноцепочечной ДНК.
[0222] В качестве линейных двухцепочечных связанных фрагментов ДНК использовали DCW34 - DCW43 (SEQ ID NO: 34 - SEQ ID NO: 43). В качестве белка рекомбиназы семейства RecA использовали RecA E. coli дикого типа. В качестве 3'→5'-экзонуклеазы, специфичной к линейной двухцепочечной ДНК, использовали экзонуклеазу III, а в качестве 3'→5'-экзонуклеазы, специфичной к одноцепочечной ДНК, использовали экзонуклеазу I и экзонуклеазу Т.
[0223] В частности, сначала приготавливали реакционные растворы, состоящие из 1 нМ каждого из DCW34-DCW43, 1 мкМ RecA дикого типа, 80 мЕд/мкл экзонуклеазы III, 1 ед./мкл экзонуклеазы I, 20 мМ Триса-HCl (рН 8,0), 4 мМ DТТ, 1 мМ ацетата магния, 50 мМ глутамата калия, 100 мкМ АТФ, 150 мМ ТМАС, 5% по массе ПЭГ8000, 10% по объему ДМСО, 20 нг/мкл креатинкиназы, 4 мМ фосфата креатина и 0 ед./мкл (без добавления), 0,05 ед./мкл, 0,15 ед./мкл или 0,5 ед./мкл экзонуклеазы Т. Затем эти реакционные растворы инкубировали при 42°C в течение 30 минут для проведения реакции связывания, а затем инкубировали при 65°C в течение 2 минут для термообработки, после чего быстро охлаждали на льду. После термообработки и быстрого охлаждения, 1,5 мкл реакционных растворов подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0224] Результаты окрашивания представлены на фиг. 27. В результате, во всех образцах, подвергнутых реакции связывания, наблюдалась полоса связанной молекулы, полученной путем связывания фрагментов всех 10 типов. Количество связанных молекул из 2-9 фрагментов снижалось в зависимости от содержания экзонуклеазы Т. На основании этих результатов было обнаружено, что добавление экзонуклеазы I и экзонуклеазы T стимулировало реакцию связывания множества связывающихся фрагментов.
[0225] [Пример 24]
Линейные двухцепочечные фрагменты ДНК двух или более типов были связаны с использованием белка рекомбиназы семейства RecA и 3'→5'-экзонуклеазы. В качестве белка рекомбиназы семейства RecA был использован бактериофаг RecA, который является гомологом фага Т4 UvsX. В качестве линейных двухцепочечных связанных фрагментов ДНК использовали DCW34 - DCW43 (SEQ ID NO: 34 - SEQ ID NO: 43).
[0226] В частности, сначала приготавливали реакционные растворы, состоящие из 1 нМ каждого из DCW34-DCW43, 8 мЕд/мкл, 30 мЕд/мкл или 80 мЕд/мкл экзонуклеазы III, 1 ед./мкл экзонуклеазы I, 20 мМ Триса-HCl (рН 8,0), 4 мМ DТТ, 1 мМ ацетата магния, 50 мМ глутамата калия, 100 мкМ АТФ, 150 мМ ТМАС, 5% по массе ПЭГ8000, 10% по объему ДМСО, 20 нг/мкл креатинкиназы, 4 мМ фосфата креатина и 0 ед./мкл (без добавления), 1 мкМ или 3 мкМ UvsX или 1 мкМ RecA дикого типа (контроля). Затем эти реакционные растворы инкубировали при 42°C в течение 30 минут для проведения реакции связывания, а затем инкубировали при 65°C в течение 2 минут для термообработки, после чего быстро охлаждали на льду. После термообработки и быстрого охлаждения, 1,5 мкл реакционных растворов подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0227] Результаты окрашивания представлены на фиг. 28. На этой фигуре, «Вход» означает дорожку, где используется 2 мкл раствора, содержащего 1 нМ каждого из DCW34-DCW43. В результате была детектирована полоса связанной молекулы, полученной путем связывания фрагментов всех 10 типов в образцах, полученных после реакции связывания в присутствии 1 мкМ или 3 мкМ UvsX и 80 мЕд/мкл экзонуклеазы III, где указанные образцы были аналогичны образцам, полученным после реакции связывания в присутствии 1 мкМ RecA дикого типа и 80 мЕд/мкл экзонуклеазы III. Эти результаты подтвердили, что реакция связывания может быть осуществлена с высокой эффективностью связывания даже при использовании UvsX, который представляет собой гомолог бактериофага RecA, в отличие от RecA дикого типа.
[0228] [Пример 25]
В реакции связывания линейных двухцепочечных фрагментов ДНК двух или более типов с использованием UvsX и 3'→5'-экзонуклеазы оценивали влияние использования фага Т4 вместе с UvsY. В качестве линейных двухцепочечных связанных фрагментов ДНК использовали DCW34 - DCW43 (SEQ ID NO: 34 - SEQ ID NO: 43).
[0229] В частности, сначала приготавливали реакционные растворы, состоящие из 1 нМ каждого из DCW34-DCW43, 3 мкМ UvsX, 60 мЕд/мкл экзонуклеазы III, 1 ед./мкл экзонуклеазы I, 20 мМ Триса-HCl (рН 8,0), 4 мМ DТТ, 1 мМ ацетата магния, 50 мМ глутамата калия, 100 мкМ АТФ, 150 мМ ТМАС, 5% по массе ПЭГ8000, 10% по объему ДМСО, 20 нг/мкл креатинкиназы, 4 мМ фосфата креатина и 0 ед./мкл (без добавления), 0,1 мкМ, 0,3 мкМ или 1 мкМ UvsY. Затем, эти реакционные растворы инкубировали при 42°C в течение 30 минут для проведения реакции связывания, а затем инкубировали при 65°C в течение 2 минут для термообработки, после чего быстро охлаждали на льду. После термообработки и быстрого охлаждения, 1,5 мкл реакционных растворов подвергали электрофорезу в агарозном геле, и разделенные полосы окрашивали зеленым SYBR.
[0230] Результаты окрашивания представлены на фиг. 29. В результате, во всех образцах, подвергнутых реакции связывания, была детектирована полоса связанной молекулы, полученной путем связывания фрагментов всех 10 типов. В зависимости от содержания UvsY, количество связанной молекулы, полученной путем связывания фрагментов всех 10 типов, повышалось, а количество связанных молекул из 2-9 фрагментов снижалось. Эти результаты показали, что с использованием UvsX и UvsY, взятых вместе, можно стимулировать реакцию связывания с использованием экзонуклеазы III и стимулятора UvsX.
Объяснение символов
[0231] 1a, 1b … линейный двухцепочечный фрагмент ДНК, H … гомологичная область, 2 … 3'→5' экзонуклеаза, 3 … белок рекомбиназы семейства RecA.
[Список последовательностей]
--->
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Japan Science and Technology Agency
<120> PC-25879
<130> СПОСОБ ПОЛУЧЕНИЯ ДНК И НАБОР ДЛЯ СВЯЗЫВАНИЯ ФРАГМЕНТОВ ДНК
<160> 67
<210> 1
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW1
<400> 1
ttctaaaacc gtgactgcgg atatcccgat tgtgggggat gctcgccagg tcctcgaaca 60
aatgcttgaa ctcttgtcgc aagaatccgc ccatcaacca ctggatgaga tccgcgactg 120
gtggcagcaa attgaacagt ggcgcgctcg tcagtgcctg aaatatgaca ctcacagtga 180
aaagattaaa ccgcaggcgg tgatcgagac tctttggcgg ttgacgaagg gagacgctta 240
cgtgacgtcc gatgtcgggc agcaccagat gtttgctgca ctttattatc cattcgacaa 300
accgcgtcgc tggatcaatt ccggtggcct cggcacgatg ggttttggtt tacctgcggc 360
actgggcgtc aaaatggcgt tgccagaaga aaccgtggtt tgcgtcactg gcgacggcag 420
tattcagatg aacatccagg aactgtctac cgcgttgcaa tacgagttgc ccgtactggt 480
ggtgaatctc aataaccgct atctggggat ggtgaagcag tggcaggaca tgatctattc 540
cggccgtcat tcacaatctt atatgcaatc gctacccgat ttcgtccgtc t 591
<210> 2
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW2
<400> 2
gatctattcc ggccgtcatt cacaatctta tatgcaatcg ctacccgatt tcgtccgtct 60
ggcggaagcc tatgggcatg tcgggatcca gatttctcat ccgcatgagc tggaaagcaa 120
acttagcgag gcgctggaac aggtgcgcaa taatcgcctg gtgtttgttg atgttaccgt 180
cgatggcagc gagcacgtct acccgatgca gattcgcggg ggcggaatgg atgaaatgtg 240
gttaagcaaa acggagagaa cctgattatg cgccggatat tatcagtctt actcgaaaat 300
gaatcaggcg cgttatcccg cgtgattggc cttttttccc agcgtggcta caacattgaa 360
agcctgaccg ttgcgccaac cgacgatccg acattatcgc gtatgaccat ccagaccgtg 420
ggcgatgaaa aagtacttga gcagatcgaa aagcaattac acaaactggt cgatgtcttg 480
cgcgtgagtg agttggggca gggcgcgcat gttgagcggg aaatcatgct ggtgaaaatt 540
caggccagcg gttacgggcg tgacgaagtg aaacgtaata cggaaatatt c 591
<210> 3
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW3
<400> 3
gtgaaaattc aggccagcgg ttacgggcgt gacgaagtga aacgtaatac ggaaatattc 60
cgtgggcaaa ttatcgatgt cacaccctcg ctttataccg ttcaattagc aggcaccagc 120
ggtaagcttg atgcattttt agcatcgatt cgcgatgtgg cgaaaattgt ggaggttgct 180
cgctctggtg tggtcggact ttcgcgcggc gataaaataa tgcgttgaga atgatctcaa 240
tgcgcaattt acagcccaac atgtcacgtt gggctttttt tgcgaaatca gtgggaacct 300
ggaataaaag cagttgccgc agttaatttt ctgcgcttag atgttaatga atttaaccca 360
taccagtaca atggctatgg tttttacatt ttacgcaagg ggcaattgtg aaactggatg 420
aaatcgctcg gctggcggga gtgtcgcgga ccactgcaag ctatgttatt aacggcaaag 480
cgaagcaata ccgtgtgagc gacaaaaccg ttgaaaaagt catggctgtg gtgcgtgagc 540
acaattacca cccgaacgcc gtggcagctg ggcttcgtgc tggacgcaca c 591
<210> 4
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW4
<400> 4
tgcgtgagca caattaccac ccgaacgccg tggcagctgg gcttcgtgct ggacgcacac 60
gttctattgg tcttgtgatc cccgatctgg agaacaccag ctatacccgc atcgctaact 120
atcttgaacg ccaggcgcgg caacggggtt atcaactgct gattgcctgc tcagaagatc 180
agccagacaa cgaaatgcgg tgcattgagc accttttaca gcgtcaggtt gatgccatta 240
ttgtttcgac gtcgttgcct cctgagcatc ctttttatca acgctgggct aacgacccgt 300
tcccgattgt cgcgctggac cgcgccctcg atcgtgaaca cttcaccagc gtggttggtg 360
ccgatcagga tgatgccgaa atgctggcgg aagagttacg taagtttccc gccgagacgg 420
tgctttatct tggtgcgcta ccggagcttt ctgtcagctt cctgcgtgaa caaggtttcc 480
gtactgcctg gaaagatgat ccgcgcgaag tgcatttcct gtatgccaac agctatgagc 540
gggaggcggc tgcccagtta ttcgaaaaat ggctggaaac gcatccgatg c 591
<210> 5
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW5
<400> 5
gctatgagcg ggaggcggct gcccagttat tcgaaaaatg gctggaaacg catccgatgc 60
cgcaggcgct gttcacaacg tcgtttgcgt tgttgcaagg agtgatggat gtcacgctgc 120
gtcgcgacgg caaactgcct tctgacctgg caattgccac ctttggcgat aacgaactgc 180
tcgacttctt acagtgtccg gtgctggcag tggctcaacg tcaccgcgat gtcgcagagc 240
gtgtgctgga gattgtcctg gcaagcctgg acgaaccgcg taagccaaaa cctggtttaa 300
cgcgcattaa acgtaatctc tatcgccgcg gcgtgctcag ccgtagctaa gccgcgaaca 360
aaaatacgcg ccaggtgaat ttccctctgg cgcgtagagt acgggactgg acatcaatat 420
gcttaaagta aataagacta ttcctgacta ttattgataa atgcttttaa acccgcccgt 480
taattaactc accagctgaa attcacaata attaagtgat atcgacagcg cgtttttgca 540
ttattttgtt acatgcggcg atgaattgcc gatttaacaa acacttttct t 591
<210> 6
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW6
<400> 6
gtttttgcat tattttgtta catgcggcga tgaattgccg atttaacaaa cacttttctt 60
tgcttttgcg caaacccgct ggcatcaagc gccacacaga cgtaacaagg actgttaacc 120
ggggaagata tgtcctaaaa tgccgctcgc gtcgcaaact gacactttat atttgctgtg 180
gaaaatagtg agtcatttta aaacggtgat gacgatgagg gattttttct tacagctatt 240
cataacgtta atttgcttcg cacgttggac gtaaaataaa caacgctgat attagccgta 300
aacatcgggt tttttacctc ggtatgcctt gtgactggct tgacaagctt ttcctcagct 360
ccgtaaactc ctttcagtgg gaaattgtgg ggcaaagtgg gaataagggg tgaggctggc 420
atgttccggg gagcaacgtt agtcaatctc gacagcaaag ggcgcttatc agtgcctacc 480
cgttatcggg aacagctgct tgagaacgct gccggtcaaa tggtttgcac cattgacatt 540
tatcacccgt gcctgctgct ttaccccctg cctgaatggg aaattatcga g 591
<210> 7
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW7
<400> 7
attgacattt atcacccgtg cctgctgctt taccccctgc ctgaatggga aattatcgag 60
caaaaattat cgcgtctgtc gagcatgaac ccggttgagc gccgtgtgca gcgcctactg 120
ttaggtcatg ccagcgaatg tcagatggat ggcgcaggtc gattgttaat cgcgccagta 180
ctgcggcaac atgccgggct gacaaaagaa gtgatgctgg ttggacagtt caacaagttt 240
gagctgtggg atgaaacaac ctggcatcaa caggtcaagg aagatatcga cgcagagcag 300
ttggctaccg gagacttatc ggagcgactg caggacttgt ctctataaaa tgatggaaaa 360
ctataaacat actacggtgc tgctggatga agccgttaat ggcctcaata tccgtcctga 420
tggcatctac attgatggga cttttggtcg cggtggtcac tcacgtctga tcctctcgca 480
gcttggcgaa gaggggcgtt tgctggcgat cgatcgcgac ccgcaggcta tcgccgttgc 540
gaagactatt gatgatccgc gcttctccat catccacgga cctttctccg c 591
<210> 8
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW8
<400> 8
cgccgttgcg aagactattg atgatccgcg cttctccatc atccacggac ctttctccgc 60
gctgggcgaa tacgttgccg agcgcgatct tatcggcaag atcgacggca ttctcctcga 120
tcttggcgtc tcttcaccgc aacttgatga tgctgaacgt ggcttttcct ttatgcgcga 180
tggtccgctg gacatgcgta tggacccaac ccgtgggcag tcagccgctg aatggctaca 240
aaccgcagaa gaagccgata tcgcctgggt attgaaaacc tatggtgaag agcgttttgc 300
caaacgcatt gcccgcgcca ttgtcgagcg taaccgcgaa cagccgatga cccgcaccaa 360
agaactggcg gaagtcgtgg ctgctgcaac gccggtgaaa gataagttta aacatcccgc 420
gacccgtacc ttccaggcgg tgcgcatttg ggtaaacagt gaactggagg agatagagca 480
ggcgctaaaa agctcgctca acgtgctggc cccgggtggg cggctttcga tcatcagctt 540
ccactcgctg gaagaccgta ttgtgaaacg ttttatgcgt gaaaacagcc g 591
<210> 9
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW9
<400> 9
catcagcttc cactcgctgg aagaccgtat tgtgaaacgt tttatgcgtg aaaacagccg 60
cggtccgcaa gttccggcag ggttaccgat gactgaagag cagctcaaaa aactgggtgg 120
ccgtcagctg cgagcactag gcaagttaat gccgggcgaa gaagaggtgg ctgagaaccc 180
tcgtgcccgt agttcagttc tgcgtattgc agagaggacg aatgcatgat cagcagagtg 240
acagaagctc taagcaaagt taaaggatcg atgggaagcc acgagcgcca tgcattgcct 300
ggtgttatcg gtgacgatct tttgcgattt gggaagctgc cactctgcct gttcatttgc 360
attattttga cggcggtgac tgtggtaacc acggcgcacc atacccgttt actgaccgct 420
cagcgcgaac aactggtgct ggagcgagat gctttagaca ttgaatggcg caacctgatc 480
cttgaagaga atgcgctcgg cgaccatagc cgggtggaaa ggatcgccac ggaaaagctg 540
caaatgcagc atgttgatcc gtcacaagaa aatatcgtag tgcaaaaata a 591
<210> 10
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW10
<400> 10
gaaaagctgc aaatgcagca tgttgatccg tcacaagaaa atatcgtagt gcaaaaataa 60
ggataaacgc gacgcatgaa agcagcggcg aaaacgcaga aaccaaaacg tcaggaagaa 120
catgccaact ttatcagttg gcgttttgcg ttgttatgcg gctgtattct cctggcgctg 180
gcttttctgc tcggacgcgt agcgtggtta caagttatct ccccggatat gctggtgaaa 240
gagggcgaca tgcgttctct tcgcgttcag caagtttcca cctcccgcgg catgattact 300
gaccgttctg gtcgcccgtt agcggtgagc gtgccggtaa aagcgatttg ggctgacccg 360
aaagaagtgc atgacgctgg cggtatcagc gtcggtgacc gctggaaggc gctggctaac 420
gcgctcaata ttccgctgga tcagctttca gcccgcatta acgccaaccc gaaagggcgc 480
tttatttatc tggcgcgtca ggtgaaccct gacatggcgg actacatcaa aaaactgaaa 540
ctgccgggga ttcatctgcg tgaagagtct cgccgttact atccgtccgg c 591
<210> 11
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW11
<400> 11
aaactgaaac tgccggggat tcatctgcgt gaagagtctc gccgttacta tccgtccggc 60
gaagtgactg ctcacctcat cggctttact aacgtcgata gtcaagggat tgagggcgtt 120
gagaagagtt tcgataaatg gcttaccggg cagccgggtg agcgcattgt gcgtaaagac 180
cgctatggtc gcgtaattga agatatttct tctactgaca gccaggcagc gcacaacctg 240
gcgctgagta ttgatgaacg cctgcaggcg ctggtttatc gcgaactgaa caacgcggtg 300
gcctttaaca aggctgaatc tggtagcgcc gtgctggtgg atgtcaacac cggtgaagtg 360
ctggcgatgg ctaacagccc gtcatacaac cctaacaatc tgagcggcac gccgaaagag 420
gcgatgcgta accgtaccat caccgacgtg tttgaaccgg gctcaacggt taaaccgatg 480
gtggtaatga ccgcgttgca acgtggcgtg gtgcgggaaa actcggtact caataccatt 540
ccttatcgaa ttaacggcca cgaaatcaaa gacgtggcac gctacagcga a 591
<210> 12
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW12
<400> 12
aataccattc cttatcgaat taacggccac gaaatcaaag acgtggcacg ctacagcgaa 60
ttaaccctga ccggggtatt acagaagtcg agtaacgtcg gtgtttccaa gctggcgtta 120
gcgatgccgt cctcagcgtt agtagatact tactcacgtt ttggactggg aaaagcgacc 180
aatttggggt tggtcggaga acgcagtggc ttatatcctc aaaaacaacg gtggtctgac 240
atagagaggg ccaccttctc tttcggctac gggctaatgg taacaccatt acagttagcg 300
cgagtctacg caactatcgg cagctacggc atttatcgcc cactgtcgat taccaaagtt 360
gaccccccgg ttcccggtga acgtgtcttc ccggaatcca ttgtccgcac tgtggtgcat 420
atgatggaaa gcgtggcgct accaggcggc ggcggcgtga aggcggcgat taaaggctat 480
cgtatcgcca ttaaaaccgg taccgcgaaa aaggtcgggc cggacggtcg ctacatcaat 540
aaatatattg cttataccgc aggcgttgcg cctgcgagtc agccgcgctt c 591
<210> 13
<211> 571
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW13b
<400> 13
taaatatatt gcttataccg caggcgttgc gcctgcgagt cagccgcgct tcgcgctggt 60
tgttgttatc aacgatccgc aggcgggtaa atactacggc ggcgccgttt ccgcgccggt 120
ctttggtgcc atcatgggcg gcgtattgcg taccatgaac atcgagccgg atgcgctgac 180
aacgggcgat aaaaatgaat ttgtgattaa tcaaggcgag gggacaggtg gcagatcgta 240
atttgcgcga ccttcttgct ccgtgggtgc cagacgcacc ttcgcgagca ctgcgagaga 300
tgacactcga cagccgtgtg gctgcggcgg gcgatctctt tgtagctgta gtaggtcatc 360
aggcggacgg gcgtcgatat atcccgcagg cgatagcgca aggtgtggct gccattattg 420
cagaggcgaa agatgaggcg accgatggtg aaatccgtga aatgcacggc gtaccggtca 480
tctatctcag ccagctcaac gagcgtttat ctgcactggc gggccgcttt taccatgaac 540
cctctgacaa tttacgtctc gtgggcgtaa c 571
<210> 14
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW14
<400> 14
ccgcttttac catgaaccct ctgacaattt acgtctcgtg ggcgtaacgg gcaccaacgg 60
caaaaccacg actacccagc tgttggcgca gtggagccaa ctgcttggcg aaatcagcgc 120
ggtaatgggc accgttggta acggcctgct ggggaaagtg atcccgacag aaaatacaac 180
cggttcggca gtcgatgttc agcatgagct ggcggggctg gtggatcagg gcgcgacgtt 240
ttgcgcaatg gaagtttcct cccacgggct ggtacagcac cgtgtggcgg cattgaaatt 300
tgcggcgtcg gtctttacca acttaagccg cgatcacctt gattatcatg gtgatatgga 360
acactacgaa gccgcgaaat ggctgcttta ttctgagcat cattgcggtc aggcgattat 420
taacgccgac gatgaagtgg gccgccgctg gctggcaaaa ctgccggacg cggttgcggt 480
atcaatggaa gatcatatta atccgaactg tcacggacgc tggttgaaag cgaccgaagt 540
gaactatcac gacagcggtg cgacgattcg ctttagctca agttggggcg a 591
<210> 15
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW15
<400> 15
gaccgaagtg aactatcacg acagcggtgc gacgattcgc tttagctcaa gttggggcga 60
tggcgaaatt gaaagccatc tgatgggcgc ttttaacgtc agcaacctgc tgctcgcgct 120
ggcgacactg ttggcactcg gctatccact ggctgatctg ctgaaaaccg ccgcgcgtct 180
gcaaccggtt tgcggacgta tggaagtgtt cactgcgcca ggcaaaccga cggtggtggt 240
ggattacgcg catacgccgg atgcactgga aaaagcctta caggcggcgc gtctgcactg 300
tgcgggcaag ctgtggtgtg tctttggctg tggtggcgat cgcgataaag gtaagcgtcc 360
actgatgggc gcaattgccg aagagtttgc tgacgtggcg gtggtgacgg acgataaccc 420
gcgtaccgaa gaaccgcgtg ccatcatcaa cgatattctg gcgggaatgt tagatgccgg 480
acatgccaaa gtgatggaag gccgtgctga agcggtgact tgcgccgtta tgcaggctaa 540
agagaatgat gtggtactgg tcgcgggcaa aggccatgaa gattaccaga t 591
<210> 16
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW16
<400> 16
gcaggctaaa gagaatgatg tggtactggt cgcgggcaaa ggccatgaag attaccagat 60
tgttggcaat cagcgtctgg actactccga tcgcgtcacg gtggcgcgtc tgctgggggt 120
gattgcatga ttagcgtaac ccttagccaa cttaccgaca ttctcaacgg tgaactgcaa 180
ggtgcagata tcacccttga tgctgtaacc actgataccc gaaaactgac gccgggctgc 240
ctgtttgttg ccctgaaagg cgaacgtttt gatgcccacg attttgccga ccaggcgaaa 300
gctggcggcg caggcgcact actggttagc cgtccgctgg acatcgacct gccgcagtta 360
atcgtcaagg atacgcgtct ggcgtttggt gaactggctg catgggttcg ccagcaagtt 420
ccggcgcgcg tggttgctct gacggggtcc tccggcaaaa cctccgttaa agagatgacg 480
gcggcgattt taagccagtg cggcaacacg ctttatacgg caggcaatct caacaacgac 540
atcggtgtac cgatgacgct gttgcgctta acgccggaat acgattacgc a 591
<210> 17
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW17
<400> 17
aacaacgaca tcggtgtacc gatgacgctg ttgcgcttaa cgccggaata cgattacgca 60
gttattgaac ttggcgcgaa ccatcagggc gaaatagcct ggactgtgag tctgactcgc 120
ccggaagctg cgctggtcaa caacctggca gcggcgcatc tggaaggttt tggctcgctt 180
gcgggtgtcg cgaaagcgaa aggtgaaatc tttagcggcc tgccggaaaa cggtatcgcc 240
attatgaacg ccgacaacaa cgactggctg aactggcaga gcgtaattgg ctcacgcaaa 300
gtgtggcgtt tctcacccaa tgccgccaac agcgatttca ccgccaccaa tatccatgtg 360
acctcgcacg gtacggaatt taccctacaa accccaaccg gtagcgtcga tgttctgctg 420
ccgttgccgg ggcgtcacaa tattgcgaat gcgctggcag ccgctgcgct ctccatgtcc 480
gtgggcgcaa cgcttgatgc tatcaaagcg gggctggcaa atctgaaagc tgttccaggc 540
cgtctgttcc ccatccaact ggcagaaaac cagttgctgc tcgacgactc c 591
<210> 18
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW18
<400> 18
gttccaggcc gtctgttccc catccaactg gcagaaaacc agttgctgct cgacgactcc 60
tacaacgcca atgtcggttc aatgactgca gcagtccagg tactggctga aatgccgggc 120
taccgcgtgc tggtggtggg cgatatggcg gaactgggcg ctgaaagcga agcctgccat 180
gtacaggtgg gcgaggcggc aaaagctgct ggtattgacc gcgtgttaag cgtgggtaaa 240
caaagccatg ctatcagcac cgccagcggc gttggcgaac attttgctga taaaactgcg 300
ttaattacgc gtcttaaatt actgattgct gagcaacagg taattacgat tttagttaag 360
ggttcacgta gtgccgccat ggaagaggta gtacgcgctt tacaggagaa tgggacatgt 420
tagtttggct ggccgaacat ttggtcaaat attattccgg ctttaacgtc ttttcctatc 480
tgacgtttcg cgccatcgtc agcctgctga ccgcgctgtt catctcattg tggatgggcc 540
cgcgtatgat tgctcatttg caaaaacttt cctttggtca ggtggtgcgt a 591
<210> 19
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW19
<400> 19
ggatgggccc gcgtatgatt gctcatttgc aaaaactttc ctttggtcag gtggtgcgta 60
acgacggtcc tgaatcacac ttcagcaagc gcggtacgcc gaccatgggc gggattatga 120
tcctgacggc gattgtgatc tccgtactgc tgtgggctta cccgtccaat ccgtacgtct 180
ggtgcgtgtt ggtggtgctg gtaggttacg gtgttattgg ctttgttgat gattatcgca 240
aagtggtgcg taaagacacc aaagggttga tcgctcgttg gaagtatttc tggatgtcgg 300
tcattgcgct gggtgtcgcc ttcgccctgt accttgccgg caaagacacg cccgcaacgc 360
agctggtggt cccattcttt aaagatgtga tgccgcagct ggggctgttc tacattctgc 420
tggcttactt cgtcattgtg ggtactggca acgcggtaaa cctgaccgat ggtctcgacg 480
gcctggcaat tatgccgacc gtatttgtcg ccggtggttt tgcgctggtg gcgtgggcga 540
ccggcaatat gaactttgcc agctacttgc atataccgta tctgcgacac g 591
<210> 20
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW20
<400> 20
cgtgggcgac cggcaatatg aactttgcca gctacttgca tataccgtat ctgcgacacg 60
ccggggaact ggttattgtc tgtaccgcga tagtcggggc aggactgggc ttcctgtggt 120
ttaacaccta tccggcgcag gtctttatgg gcgatgtagg ttcgctggcg ttaggtggtg 180
cgttaggcat tatcgccgta ctgctacgtc aggaattcct gctggtgatt atggggggcg 240
tgttcgtggt agaaacgctt tctgtcatcc tgcaggtcgg ctcctttaaa ctgcgcggac 300
aacgtatttt ccgcatggca ccgattcatc accactatga actgaaaggc tggccggaac 360
cgcgcgtcat tgtgcgtttc tggattattt cgctgatgct ggttctgatt ggtctggcaa 420
cgctgaaggt acgttaatca tggctgatta tcagggtaaa aatgtcgtca ttatcggcct 480
gggcctcacc gggctttcct gcgtggactt tttcctcgct cgcggtgtga cgccgcgcgt 540
tatggatacg cgtatgacac cgcctggcct ggataaatta cccgaagccg t 591
<210> 21
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW21
<400> 21
gccgcgcgtt atggatacgc gtatgacacc gcctggcctg gataaattac ccgaagccgt 60
agaacgccac acgggcagtc tgaatgatga atggctgatg gcggcagatc tgattgtcgc 120
cagtcccggt attgcactgg cgcatccatc cttaagcgct gccgctgatg ccggaatcga 180
aatcgttggc gatatcgagc tgttctgtcg cgaagcacaa gcaccgattg tggcgattac 240
cggttctaac ggcaaaagca cggtcaccac gctagtgggt gaaatggcga aagcggcggg 300
ggttaacgtt ggtgtgggtg gcaatattgg cctgcctgcg ttgatgctac tggatgatga 360
gtgtgaactg tacgtgctgg aactgtcgag cttccagctg gaaaccacct ccagcttaca 420
ggcggtagca gcgaccattc tgaacgtgac tgaagatcat atggatcgct atccgtttgg 480
tttacaacag tatcgtgcag caaaactgcg catttacgaa aacgcgaaag tttgcgtggt 540
taatgctgat gatgccttaa caatgccgat tcgcggtgcg gatgaacgct g 591
<210> 22
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW22
<400> 22
ttgcgtggtt aatgctgatg atgccttaac aatgccgatt cgcggtgcgg atgaacgctg 60
cgtcagcttt ggcgtcaaca tgggtgacta tcacctgaat catcagcagg gcgaaacctg 120
gctgcgggtt aaaggcgaga aagtgctgaa tgtgaaagag atgaaacttt ccgggcagca 180
taactacacc aatgcgctgg cggcgctggc gctggcagat gctgcagggt taccgcgtgc 240
cagcagcctg aaagcgttaa ccacattcac tggtctgccg catcgctttg aagttgtgct 300
ggagcataac ggcgtacgtt ggattaacga ttcgaaagcg accaacgtcg gcagtacgga 360
agcggcgctg aatggcctgc acgtagacgg cacactgcat ttgttgctgg gtggcgatgg 420
taaatcggcg gactttagcc cactggcgcg ttacctgaat ggcgataacg tacgtctgta 480
ttgtttcggt cgtgacggcg cgcagctggc ggcgctacgc ccggaagtgg cagaacaaac 540
cgaaactatg gaacaggcga tgcgcttgct ggctccgcgt gttcagccgg g 591
<210> 23
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW23
<400> 23
agaacaaacc gaaactatgg aacaggcgat gcgcttgctg gctccgcgtg ttcagccggg 60
cgatatggtt ctgctctccc cagcctgtgc cagccttgat cagttcaaga actttgaaca 120
acgaggcaat gagtttgccc gtctggcgaa ggagttaggt tgatgcgttt atctctccct 180
cgcctgaaaa tgccgcgcct gccaggattc agtatcctgg tctggatctc cacggcgcta 240
aagggctggg tgatgggctc gcgggaaaaa gataccgaca gcctgatcat gtacgatcgc 300
accttactgt ggctgacctt cggcctcgcg gcgattggct ttatcatggt gacctcggcg 360
tcaatgccca tagggcaacg cttaaccaac gatccgttct tcttcgcgaa gcgtgatggt 420
gtctatctga ttttggcgtt tattctggcg atcattacgc tgcgtctgcc gatggagttc 480
tggcaacgct acagtgccac gatgctgctc ggatctatca tcctgctgat gatcgtcctg 540
gtagtgggta gctcggttaa aggggcatcg cgttggatcg atctcggttt g 591
<210> 24
<211> 589
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW24b
<400> 24
atcgtcctgg tagtgggtag ctcggttaaa ggggcatcgc gttggatcga tctcggtttg 60
ctgcgtatcc agcctgcgga gctgacaaaa ctgtcgctgt tttgctatat cgccaactat 120
ctggtgcgta aaggcgacga agtacgtaat aacctgcgcg gcttcctgaa accgatgggc 180
gtgattctgg tgttggcagt gttactgctg gcacagccag accttggtac ggtggtggtg 240
ttgtttgtga ctacgctggc gatgttgttc ctggcgggag cgaaattgtg gcagttcatt 300
gccattatcg gtatgggcat ttcagcggtt gtgttgctga tactcgccga accgtaccgt 360
atccgccgtg ttaccgcatt ctggaacccg tgggaagatc cctttggcag cggctatcag 420
ttaacgcaat cgctgatggc gtttggtcgc ggcgaacttt gggggcaagg tttaggtaac 480
tcggtacaaa aactggagta tctgccggaa gcgcacactg actttatttt cgccattatc 540
ggcgaagaac tggggtatgt cggtgtggtg ctggcacttt taatggtat 589
<210> 25
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW25
<400> 25
gccattatcg gcgaagaact ggggtatgtc ggtgtggtgc tggcactttt aatggtattc 60
ttcgtcgctt ttcgcgcgat gtcgattggc cgtaaagcat tagaaattga ccaccgtttt 120
tccggttttc tcgcctgttc tattggcatc tggtttagct tccaggcgct ggttaacgta 180
ggcgcggcgg cggggatgtt accgaccaaa ggtctgacat tgccgctgat cagttacggt 240
ggttcgagct tactgattat gtcgacagcc atcatgatgc tgttgcgtat tgattatgaa 300
acgcgtctgg agaaagcgca ggcgtttgta cgaggttcac gatgagtggt caaggaaagc 360
gattaatggt gatggcaggc ggaaccggtg gacatgtatt cccgggactg gcggttgcgc 420
accatctaat ggctcagggt tggcaagttc gctggctggg gactgccgac cgtatggaag 480
cggacttagt gccaaaacat ggcatcgaaa ttgatttcat tcgtatctct ggtctgcgtg 540
gaaaaggtat aaaagcactg atagctgccc cgctgcgtat cttcaacgcc t 591
<210> 26
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW26
<400> 26
gtctgcgtgg aaaaggtata aaagcactga tagctgcccc gctgcgtatc ttcaacgcct 60
ggcgtcaggc gcgggcgatt atgaaagcgt acaaacctga cgtggtgctc ggtatgggag 120
gctacgtgtc aggtccaggt ggtctggccg cgtggtcgtt aggcattccg gttgtacttc 180
atgaacaaaa cggtattgcg ggcttaacca ataaatggct ggcgaagatt gccaccaaag 240
tgatgcaggc gtttccaggt gctttcccta atgcggaagt agtgggtaac ccggtgcgta 300
ccgatgtgtt ggcgctgccg ttgccgcagc aacgtttggc tggacgtgaa ggtccggttc 360
gtgtgctggt agtgggtggt tctcagggcg cacgcattct taaccagaca atgccgcagg 420
ttgctgcgaa actgggtgat tcagtcacta tctggcatca gagcggcaaa ggttcgcaac 480
aatccgttga acaggcgtat gccgaagcgg ggcaaccgca gcataaagtg acggaattta 540
ttgatgatat ggcggcggcg tatgcgtggg cggatgtcgt cgtttgccgc t 591
<210> 27
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW27
<400> 27
cggaatttat tgatgatatg gcggcggcgt atgcgtgggc ggatgtcgtc gtttgccgct 60
ccggtgcgtt aacggtgagt gaaatcgccg cggcaggact accggcgttg tttgtgccgt 120
ttcaacataa agaccgccag caatactgga atgcgctacc gctggaaaaa gcgggcgcag 180
ccaaaattat cgagcagcca cagcttagcg tggatgctgt cgccaacacc ctggccgggt 240
ggtcgcgaga aaccttatta accatggcag aacgcgcccg cgctgcatcc attccggatg 300
ccaccgagcg agtggcaaat gaagtgagcc gggttgcccg ggcgtaattg tagcgatgcc 360
ttttgcatcg tatgaattta agaagttaat ggcgtaaaga atgaatacac aacaattggc 420
aaaactgcgt tccatcgtgc ccgaaatgcg tcgcgttcgg cacatacatt ttgtcggcat 480
tggtggtgcc ggtatgggcg gtattgccga agttctggcc aatgaaggtt atcagatcag 540
tggttccgat ttagcgccaa atccggtcac gcagcagtta atgaatctgg g 591
<210> 28
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW28
<400> 28
tcagatcagt ggttccgatt tagcgccaaa tccggtcacg cagcagttaa tgaatctggg 60
tgcgacgatt tatttcaacc atcgcccgga aaacgtacgt gatgccagcg tggtcgttgt 120
ttccagcgcg atttctgccg ataacccgga aattgtcgcc gctcatgaag cgcgtattcc 180
ggtgatccgt cgtgccgaaa tgctggctga gttaatgcgt tttcgtcatg gcatcgccat 240
tgccggaacg cacggcaaaa cgacaaccac cgcgatggtt tccagcatct acgcagaagc 300
ggggctcgac ccaaccttcg ttaacggcgg gctggtaaaa gcggcggggg ttcatgcgcg 360
tttggggcat ggtcggtacc tgattgccga agcagatgag agtgatgcat cgttcctgca 420
tctgcaaccg atggtggcga ttgtcaccaa tatcgaagcc gaccacatgg atacctacca 480
gggcgacttt gagaatttaa aacagacttt tattaatttt ctgcacaacc tgccgtttta 540
cggtcgtgcg gtgatgtgtg ttgatgatcc ggtgatccgc gaattgttac c 591
<210> 29
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW29
<400> 29
gccgttttac ggtcgtgcgg tgatgtgtgt tgatgatccg gtgatccgcg aattgttacc 60
gcgagtgggg cgtcagacca cgacttacgg cttcagcgaa gatgccgacg tgcgtgtaga 120
agattatcag cagattggcc cgcaggggca ctttacgctg ctgcgccagg acaaagagcc 180
gatgcgcgtc accctgaatg cgccaggtcg tcataacgcg ctgaacgccg cagctgcggt 240
tgcggttgct acggaagagg gcattgacga cgaggctatt ttgcgggcgc ttgaaagctt 300
ccaggggact ggtcgccgtt ttgatttcct cggtgaattc ccgctggagc cagtgaatgg 360
taaaagcggt acggcaatgc tggtcgatga ctacggccac cacccgacgg aagtggacgc 420
caccattaaa gcggcgcgcg caggctggcc ggataaaaac ctggtaatgc tgtttcagcc 480
gcaccgtttt acccgtacgc gcgacctgta tgatgatttc gccaatgtgc tgacgcaggt 540
tgataccctg ttgatgctgg aagtgtatcc ggctggcgaa gcgccaattc c 591
<210> 30
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW30
<400> 30
gacgcaggtt gataccctgt tgatgctgga agtgtatccg gctggcgaag cgccaattcc 60
gggagcggac agccgttcgc tgtgtcgcac aattcgtgga cgtgggaaaa ttgatcccat 120
tctggtgccg gatccggcgc gggtagccga gatgctggca ccggtattaa ccggtaacga 180
cctgattctc gttcaggggg ctggtaatat tggaaaaatt gcccgttctt tagctgaaat 240
caaactgaag ccgcaaactc cggaggaaga acaacatgac tgataaaatc gcggtcctgt 300
tgggtgggac ctccgctgag cgggaagttt ctctgaattc tggcgcagcg gtgttagccg 360
gactgcgtga aggcggtatt gacgcgtatc ctgtcgaccc gaaagaagtc gacgtgacgc 420
aactgaagtc gatgggcttt cagaaagtgt ttatcgcgct acacggtcgc ggcggtgaag 480
atggtacgct gcaggggatg ctcgagctga tgggcttgcc ttataccgga agcggagtga 540
tggcatctgc gctttcaatg gataaactac gcagcaaact tctatggcaa g 591
<210> 31
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW31
<400> 31
gcggagtgat ggcatctgcg ctttcaatgg ataaactacg cagcaaactt ctatggcaag 60
gtgccggttt accggtcgcg ccgtgggtag cgttaacccg cgcagagttt gaaaaaggcc 120
tgagcgataa gcagttagca gaaatttctg ctctgggttt gccggttatc gttaagccga 180
gccgcgaagg ttccagtgtg ggaatgtcaa aagtagtagc agaaaatgct ctacaagatg 240
cattaagatt ggcatttcag cacgatgaag aagtattgat tgaaaaatgg ctaagtgggc 300
cggagttcac ggttgcgata ctcggtgaag aaattttacc gtcaatacgt attcaaccgt 360
ccggaacctt ctatgattat gaggcgaagt atctctctga tgagacacag tatttctgcc 420
ccgcaggtct ggaagcgtca caagaggcca atttgcaggc attagtgctg aaagcatgga 480
cgacgttagg ttgcaaagga tggggacgta ttgacgttat gctggacagc gatggacagt 540
tttatctgct ggaagccaat acctcaccgg gtatgaccag ccacagcctg g 591
<210> 32
<211> 579
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW32b
<400> 32
atggacagtt ttatctgctg gaagccaata cctcaccggg tatgaccagc cacagcctgg 60
tgccgatggc ggcacgtcag gcaggtatga gcttctcgca gttggtagta cgaattctgg 120
aactggcgga ctaatatgtc gcaggctgct ctgaacacgc gaaacagcga agaagaggtt 180
tcttctcgcc gcaataatgg aacgcgtctg gcggggatcc ttttcctgct gaccgtttta 240
acgacagtgt tggtgagcgg ctgggtcgtg ttgggctgga tggaagatgc gcaacgcctg 300
ccgctctcaa agctggtgtt gaccggtgaa cgccattaca cacgtaatga cgatatccgg 360
cagtcgatcc tggcattggg tgagccgggt acctttatga cccaggatgt caacatcatc 420
cagacgcaaa tagaacaacg cctgccgtgg attaagcagg tgagcgtcag aaagcagtgg 480
cctgatgaat tgaagattca tctggttgaa tatgtgccga ttgcgcggtg gaatgatcaa 540
catatggtag acgcggaagg aaataccttc agcgtgccg 579
<210> 33
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW33
<400> 33
aatgatcaac atatggtaga cgcggaagga aataccttca gcgtgccgcc agaacgcacc 60
agcaagcagg tgcttccaat gctgtatggc ccggaaggca gcgccaatga agtgttgcag 120
ggctatcgcg aaatggggca gatgctggca aaggacagat ttactctgaa ggaagcggcg 180
atgaccgcgc ggcgttcctg gcagttgacg ctgaataacg atattaagct caatcttggc 240
cggggcgata cgatgaaacg tttggctcgc tttgtagaac tttatccggt tttacagcag 300
caggcgcaaa ccgatggcaa acggattagc tacgttgatt tgcgttatga ctctggagcg 360
gcagtaggct gggcgccctt gccgccagag gaatctactc agcaacaaaa tcaggcacag 420
gcagaacaac aatgatcaag gcgacggaca gaaaactggt agtaggactg gagattggta 480
ccgcgaaggt tgccgcttta gtaggggaag ttctgcccga cggtatggtc aatatcattg 540
gcgtgggcag ctgcccgtcg cgtggtatgg ataaaggcgg ggtgaacgac c 591
<210> 34
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW34
<400> 34
atatcattgg cgtgggcagc tgcccgtcgc gtggtatgga taaaggcggg gtgaacgacc 60
tcgaatccgt ggtcaagtgc gtacaacgcg ccattgacca ggcagaattg atggcagatt 120
gtcagatctc ttcggtatat ctggcgcttt ctggtaagca catcagctgc cagaatgaaa 180
ttggtatggt gcctatttct gaagaagaag tgacgcaaga agatgtggaa aacgtcgtcc 240
ataccgcgaa atcggtgcgt gtgcgcgatg agcatcgtgt gctgcatgtg atcccgcaag 300
agtatgcgat tgactatcag gaagggatca agaatccggt aggactttcg ggcgtgcgga 360
tgcaggcaaa agtgcacctg atcacatgtc acaacgatat ggcgaaaaac atcgtcaaag 420
cggttgaacg ttgtgggctg aaagttgacc aactgatatt tgccggactg gcatcaagtt 480
attcggtatt gacggaagat gaacgtgaac tgggtgtctg cgtcgtcgat atcggtggtg 540
gtacaatgga tatcgccgtt tataccggtg gggcattgcg ccacactaag g 591
<210> 35
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW35
<400> 35
tcggtggtgg tacaatggat atcgccgttt ataccggtgg ggcattgcgc cacactaagg 60
taattcctta tgctggcaat gtcgtgacca gtgatatcgc ttacgccttt ggcacgccgc 120
caagcgacgc cgaagcgatt aaagttcgcc acggttgtgc gctgggttcc atcgttggaa 180
aagatgagag cgtggaagtg ccgagcgtag gtggtcgtcc gccacggagt ctgcaacgtc 240
agacactggc agaggtgatc gagccgcgct ataccgagct gctcaacctg gtcaacgaag 300
agatattgca gttgcaggaa aagcttcgcc aacaaggggt taaacatcac ctggcggcag 360
gcattgtatt aaccggtggc gcagcgcaga tcgaaggtct tgcagcctgt gctcagcgcg 420
tgtttcatac gcaagtgcgt atcggcgcgc cgctgaacat taccggttta acggattatg 480
ctcaggagcc gtattattcg acggcggtgg gattgcttca ctatgggaaa gagtcacatc 540
ttaacggtga agctgaagta gaaaaacgtg ttacagcatc agttggctcg t 591
<210> 36
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW36
<400> 36
agtcacatct taacggtgaa gctgaagtag aaaaacgtgt tacagcatca gttggctcgt 60
ggatcaagcg actcaatagt tggctgcgaa aagagtttta atttttatga ggccgacgat 120
gattacggcc tcaggcgaca ggcacaaatc ggagagaaac tatgtttgaa ccaatggaac 180
ttaccaatga cgcggtgatt aaagtcatcg gcgtcggcgg cggcggcggt aatgctgttg 240
aacacatggt gcgcgagcgc attgaaggtg ttgaattctt cgcggtaaat accgatgcac 300
aagcgctgcg taaaacagcg gttggacaga cgattcaaat cggtagcggt atcaccaaag 360
gactgggcgc tggcgctaat ccagaagttg gccgcaatgc ggctgatgag gatcgcgatg 420
cattgcgtgc ggcgctggaa ggtgcagaca tggtctttat tgctgcgggt atgggtggtg 480
gtaccggtac aggtgcagca ccagtcgtcg ctgaagtggc aaaagatttg ggtatcctga 540
ccgttgctgt cgtcactaag cctttcaact ttgaaggcaa gaagcgtatg g 591
<210> 37
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW37
<400> 37
gtatcctgac cgttgctgtc gtcactaagc ctttcaactt tgaaggcaag aagcgtatgg 60
cattcgcgga gcaggggatc actgaactgt ccaagcatgt ggactctctg atcactatcc 120
cgaacgacaa actgctgaaa gttctgggcc gcggtatctc cctgctggat gcgtttggcg 180
cagcgaacga tgtactgaaa ggcgctgtgc aaggtatcgc tgaactgatt actcgtccgg 240
gtttgatgaa cgtggacttt gcagacgtac gcaccgtaat gtctgagatg ggctacgcaa 300
tgatgggttc tggcgtggcg agcggtgaag accgtgcgga agaagctgct gaaatggcta 360
tctcttctcc gctgctggaa gatatcgacc tgtctggcgc gcgcggcgtg ctggttaaca 420
tcacggcggg cttcgacctg cgtctggatg agttcgaaac ggtaggtaac accatccgtg 480
catttgcttc cgacaacgcg actgtggtta tcggtacttc tcttgacccg gatatgaatg 540
acgagctgcg cgtaaccgtt gttgcgacag gtatcggcat ggacaaacgt c 591
<210> 38
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW38
<400> 38
atatgaatga cgagctgcgc gtaaccgttg ttgcgacagg tatcggcatg gacaaacgtc 60
ctgaaatcac tctggtgacc aataagcagg ttcagcagcc agtgatggat cgctaccagc 120
agcatgggat ggctccgctg acccaggagc agaagccggt tgctaaagtc gtgaatgaca 180
atgcgccgca aactgcgaaa gagccggatt atctggatat cccagcattc ctgcgtaagc 240
aagctgatta agaattgact ggaatttggg tttcgaggct ctttgtgcta aactggcccg 300
ccgaatgtat agtacacttc ggttggatag gtaatttggc gagataatac gatgatcaaa 360
caaaggacac ttaaacgtat cgttcaggcg acgggtgtcg gtttacatac cggcaagaaa 420
gtcaccctga cgttacgccc tgcgccggcc aacaccgggg tcatctatcg tcgcaccgac 480
ttgaatccac cggtagattt cccggccgat gccaaatctg tgcgtgatac catgctctgt 540
acgtgtctgg tcaacgagca tgatgtacgg atttcaaccg tagagcacct c 591
<210> 39
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW39
<400> 39
atgctctgta cgtgtctggt caacgagcat gatgtacgga tttcaaccgt agagcacctc 60
aatgctgctc tcgcgggctt gggcatcgat aacattgtta tcgaagttaa cgcgccggaa 120
atcccgatca tggacggcag cgccgctccg tttgtatacc tgctgcttga cgccggtatc 180
gacgagttga actgcgccaa aaaatttgtt cgcatcaaag agactgttcg tgtcgaagat 240
ggcgataagt gggctgaatt taagccgtac aatggttttt cgctggattt caccatcgat 300
tttaaccatc cggctattga ttccagcaac cagcgctatg cgatgaactt ctccgctgat 360
gcgtttatgc gccagatcag ccgtgcgcgt acgttcggtt tcatgcgtga tatcgaatat 420
ctgcagtccc gtggtttgtg cctgggcggc agcttcgatt gtgccatcgt tgttgacgat 480
tatcgcgtac tgaacgaaga cggcctgcgt tttgaagacg aatttgtgcg tcacaaaatg 540
ctcgatgcga tcggtgactt gttcatgtgt ggtcacaata ttattggtgc a 591
<210> 40
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW40
<400> 40
cacaaaatgc tcgatgcgat cggtgacttg ttcatgtgtg gtcacaatat tattggtgca 60
tttaccgctt ataaatccgg tcatgcactg aataacaaac tgctgcaggc tgtcctggcg 120
aaacaggaag cctgggaata tgtgaccttc caggacgacg cagaactgcc gttggccttc 180
aaagcgcctt cagctgtact ggcataacga catttatact gtcgtataaa attcgactgg 240
caaatctggc actctctccg gccaggtgaa ccagtcgttt ttttttgaat tttataagag 300
ctataaaaaa cggtgcgaac gctgttttct taagcacttt tccgcacaac ttatcttcat 360
tcgtgctgtg gactgcaggc tttaatgata agatttgtgc gctaaatacg tttgaatatg 420
atcgggatgg caataacgtg agtggaatac tgacgcgctg gcgacagttt ggtaaacgct 480
acttctggcc gcatctctta ttagggatgg ttgcggcgag tttaggtttg cctgcgctca 540
gcaacgccgc cgaaccaaac gcgcccgcaa aagcgacaac ccgcaaccac g 591
<210> 41
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW41
<400> 41
ctgcgctcag caacgccgcc gaaccaaacg cgcccgcaaa agcgacaacc cgcaaccacg 60
agccttcagc caaagttaac tttggtcaat tggccttgct ggaagcgaac acacgccgcc 120
cgaattcgaa ctattccgtt gattactggc atcaacatgc cattcgcacg gtaatccgtc 180
atctttcttt cgcaatggca ccgcaaacac tgcccgttgc tgaagaatct ttgcctcttc 240
aggcgcaaca tcttgcatta ctggatacgc tcagcgcgct gctgacccag gaaggcacgc 300
cgtctgaaaa gggttatcgc attgattatg cgcattttac cccacaagca aaattcagca 360
cgcccgtctg gataagccag gcgcaaggca tccgtgctgg ccctcaacgc ctcacctaac 420
aacaataaac ctttacttca ttttattaac tccgcaacgc ggggcgtttg agattttatt 480
atgctaatca aattgttaac taaagttttc ggtagtcgta acgatcgcac cctgcgccgg 540
atgcgcaaag tggtcaacat catcaatgcc atggaaccgg agatggaaaa a 591
<210> 42
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW42
<400> 42
ctgcgccgga tgcgcaaagt ggtcaacatc atcaatgcca tggaaccgga gatggaaaaa 60
ctctccgacg aagaactgaa agggaaaacc gcagagtttc gtgcacgtct ggaaaaaggc 120
gaagtgctgg aaaatctgat cccggaagct ttcgccgtgg tacgtgaggc aagtaagcgc 180
gtctttggta tgcgtcactt cgacgttcag ttactcggcg gtatggttct taacgaacgc 240
tgcatcgccg aaatgcgtac cggtgaagga aaaaccctga ccgcaacgct gcctgcttac 300
ctgaacgcac taaccggtaa aggcgtgcac gtagttaccg tcaacgacta cctggcgcaa 360
cgtgacgccg aaaacaaccg tccgctgttt gaattccttg gcctgactgt cggtatcaac 420
ctgccgggca tgccagcacc ggcaaagcgc gaagcttacg cagctgacat cacttacggt 480
acgaacaacg aatacggctt tgactacctg cgcgacaaca tggcgttcag ccctgaagaa 540
cgtgtacagc gtaaactgca ctatgcgctg gtggacgaag tggactccat c 591
<210> 43
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW43
<400> 43
cctgaagaac gtgtacagcg taaactgcac tatgcgctgg tggacgaagt ggactccatc 60
ctgatcgatg aagcgcgtac accgctgatc atttccggcc cggcagaaga cagctcggaa 120
atgtataaac gcgtgaataa aattattccg cacctgatcc gtcaggaaaa agaagactcc 180
gaaaccttcc agggcgaagg ccacttctcg gtggacgaaa aatctcgcca ggtgaacctg 240
accgaacgtg gtctggtgct gattgaagaa ctgctggtga aagagggcat catggatgaa 300
ggggagtctc tgtactctcc ggccaacatc atgctgatgc accacgtaac ggcggcgctg 360
cgcgctcatg cgctgtttac ccgtgacgtc gactacatcg ttaaagatgg tgaagttatc 420
atcgttgacg aacacaccgg tcgtaccatg cagggccgtc gctggtccga tggtctgcac 480
caggctgtgg aagcgaaaga aggtgtgcag atccagaacg aaaaccaaac gctggcttcg 540
atcaccttcc agaactactt ccgtctgtat gaaaaactgg cggggatgac c 591
<210> 44
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW44
<400> 44
ctggcttcga tcaccttcca gaactacttc cgtctgtatg aaaaactggc ggggatgacc 60
ggtactgctg ataccgaagc tttcgaattt agctcaatct acaagctgga taccgtcgtt 120
gttccgacca accgtccaat gattcgtaaa gatctgccgg acctggtcta catgactgaa 180
gcggaaaaaa ttcaggcgat cattgaagat atcaaagaac gtactgcgaa aggccagccg 240
gtgctggtgg gtactatctc catcgaaaaa tcggagctgg tgtcaaacga actgaccaaa 300
gccggtatta agcacaacgt cctgaacgcc aaattccacg ccaacgaagc ggcgattgtt 360
gctcaggcag gttatccggc tgcggtgact atcgcgacca atatggcggg tcgtggtaca 420
gatattgtgc tcggtggtag ctggcaggca gaagttgccg cgctggaaaa tccgaccgca 480
gagcaaattg aaaaaattaa agccgactgg caggtacgtc acgatgcggt actggaagca 540
ggtggcctgc atatcatcgg taccgagcgt cacgaatccc gtcgtatcga t 591
<210> 45
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW45
<400> 45
ctggaagcag gtggcctgca tatcatcggt accgagcgtc acgaatcccg tcgtatcgat 60
aaccagttgc gcggtcgttc tggtcgtcag ggggatgctg gttcttcccg tttctacctg 120
tcgatggaag atgcgctgat gcgtattttt gcttccgacc gagtatccgg catgatgcgt 180
aaactgggta tgaagccagg cgaagccatt gaacacccgt gggtgactaa agcgattgcc 240
aacgcccagc gtaaagttga aagccgtaac ttcgacattc gtaagcaact gctggaatat 300
gatgacgtgg ctaacgatca gcgtcgcgcc atttactccc agcgtaacga actgttggat 360
gtcagcgatg tgagcgaaac cattaacagc attcgtgaag atgtgttcaa agcgaccatt 420
gatgcctaca ttccaccaca gtcgctggaa gaaatgtggg atattccggg gctgcaggaa 480
cgtctgaaga acgatttcga cctcgatttg ccaattgccg agtggctgga taaagaacca 540
gaactgcatg aagagacgct gcgtgagcgc attctggcgc agtccatcga a 591
<210> 46
<211> 591
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW46
<400> 46
aaagaaccag aactgcatga agagacgctg cgtgagcgca ttctggcgca gtccatcgaa 60
gtgtatcagc gtaaagaaga agtggttggt gctgagatga tgcgtcactt cgagaaaggc 120
gtcatgctgc aaacgcttga ctccctgtgg aaagagcacc tggcagcgat ggactatctg 180
cgtcagggta tccacctgcg tggctacgca cagaaagatc cgaagcagga atacaaacgt 240
gaatcgttct ccatgtttgc agcgatgctg gagtcgttga aatatgaagt tatcagtacg 300
ctgagcaaag ttcaggtacg tatgcctgaa gaggttgagg agctggaaca acagcgtcgt 360
atggaagccg agcgtttagc gcaaatgcag cagcttagcc atcaggatga cgactctgca 420
gccgcagctg cactggcggc gcaaaccgga gagcgcaaag taggacgtaa cgatccttgc 480
ccgtgcggtt ctggtaaaaa atacaagcag tgccatggcc gcctgcaata aaagctaact 540
gttgaagtaa aaggtgcagg attctgcgcc ttttttatag gtttaagaca a 591
<210> 47
<211> 575
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW47b
<400> 47
gctaactgtt gaagtaaaag gtgcaggatt ctgcgccttt tttataggtt taagacaatg 60
aaaaagctgc aaattgcggt aggtattatt cgcaacgaga acaatgaaat ctttataacg 120
cgtcgcgcag cagatgcgca catggcgaat aaactggagt ttcccggcgg taaaattgaa 180
atgggtgaaa cgccggaaca ggcggtggtg cgtgaacttc aggaagaagt cgggattacc 240
ccccaacatt tttcgctatt tgaaaaactg gaatatgaat tcccggacag gcatataaca 300
ctgtggtttt ggctggtcga acgctgggaa ggggagccgt ggggtaaaga agggcaaccc 360
ggtgagtgga tgtcgctggt cggtcttaat gccgatgatt ttccgccagc caatgaaccg 420
gtaattgcga agcttaaacg tctgtaggtc agataaggcg ttttcgccgc atccgacatt 480
cgcacacgat gcctgatgcg acgctggcgc gtcttatcag gcctaaaggg atttctaact 540
cattgataaa tttgtttttg taggtcggat aaggc 575
<210> 48
<211> 559
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW48b
<400> 48
tttgtttttg taggtcggat aaggcgttca cgccgcatcc gacatttgca caagatgcct 60
gatgcgacgc tgtccgcgtc ttatcaggcc tacgtgcggc atcagacaaa tgtcactgct 120
ttggttcttc gctccagtca tcgctttcgg aaagatcgcc actgctgggg attcgttttt 180
cttcagcagc ccattctccg aggtcgatca gctgacaacg tttggagcaa aatggccgaa 240
acgggctgat ttcaccccac accaccgttt tcccgcaggt tgggcaattc accgtaatag 300
tttctgacat ttttactcct tagcaacagg ccagttcgaa atccagacgt tccggtacct 360
gtccgttttc agtgtccagc ggcataaaac gaatggcaaa acggctctta tgtccggaaa 420
tttgcggata aagctgtgaa tcgagcgaca gattcaggcg cagcaagtcg gcatcgccac 480
cgttatcctg ataaaaacca ttcaggctgg tttgtttacg gaagggggcc gactggcgaa 540
ttaaatccag caccatggt 559
<210> 49
<211> 558
<212> ДНК
<213> Искусственная последовательность
<220>
<223> DCW49
<400> 49
tttgtttacg gaagggggcc gactggcgaa ttaaatccag caccatggta agtgcctggg 60
tgagcgggtt caggctggca atccaggttt ctacctggct gtcgcgctgc gcctggggta 120
gatgcagcca aatgtgcaat gtaggtaaat caaagctgca acagccgcct gggatgctca 180
gtcgctgacg caccagagca atcaaacgat cttcacgcag aaattgcccg atacgcggcg 240
cggaaattaa tacgctcccc gccgctttta actgctgaat taatgcttca atacggctct 300
ggtccacgcc aggcacgcca atccaggtct ggagtttacg ttgctgccgg tcaagttctt 360
tcaacagctc agtgcggact tcgccgcgct cgaaaacatc cagtaattca ctgacattac 420
ggaagaaatg cagcgcgcca gcgtggtcaa cgatgggtaa attaacggtg agttgctgaa 480
tcaaaaactc aatgcgcagc catgtacgca ttttttcatt tagtggatgt tcaaaaagga 540
cctgggtctg cattacgg 558
<210> 50
<211> 1298
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Cm-oriC (DCW20)
<400> 50
gccgcgcgtt atggatacgc gtatgacacc gcctggcctg gataaattac ccgaagccgt 60
gtcggcttga gaaagacctg agtatgttgt aactaaaggt gcgcataatg tatattatgt 120
taaatggatc ctgggtatta aaaagaagat ctatttattt agagatctgt tctattgtga 180
tctcttatta ggatcgcact gccctgtgga taacaaggat ccggctttta agatcaacaa 240
cctggaaagg atcattaact gtgaatgatc ggtgatcctg gaccgtataa gctgggatca 300
gaatgagggg ttatacacaa ctcaaaaact gaacaacagt tgttctttgg ataactaccg 360
gttgatccaa gcttcctgac agagttatcc acagtagatc gcacgatctg tataacttcg 420
tatagcatac attatacgaa gttatcttta gttacaacat actccattct tctgccggat 480
cttcttacgc cccgccctgc cactcatcgc agtactgttg taattcatta agcattctgc 540
cgacatggaa gccatcacag acggcatgat gaacctgaat cgccagcggc atcagcacct 600
tgtcgccttg cgtataatat ttgcccatgg tgaaaacggg ggcgaagaag ttgtccatat 660
tggccacgtt taaatcaaaa ctggtgaaac tcacccaggg attggctgag acgaaaaaca 720
tattctcaat aaacccttta gggaaatagg ccaggttttc accgtaacac gccacatctt 780
gcgaatatat gtgtagaaac tgccggaaat cgtcgtggta ttcactccag agcgatgaaa 840
acgtttcagt ttgctcatgg aaaacggtgt aacaagggtg aacactatcc catatcacca 900
gctcaccgtc tttcattgcc atacggaatt ccggatgagc attcatcagg cgggcaagaa 960
tgtgaataaa ggccggataa aacttgtgct tatttttctt tacggtcttt aaaaaggccg 1020
taatatccag ctgaacggtc tggttatagg tacattgagc aactgactga aatgcctcaa 1080
aatgttcttt acgatgccat tgggatatat caacggtggt atatccagtg atttttttct 1140
ccattttagc ttccttagct cctgaaaatc tcgataactc aaaaaatacg cccggtagtg 1200
atcttatttc attatggtga aagttggaac ctcttacgtt ctaaaaccgt gactgcggat 1260
atcccgattg tgggggatgc tcgccaggtc ctcgaaca 1298
<210> 51
<211> 1298
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Cm-oriC (DCW25)
<400> 51
gtctgcgtgg aaaaggtata aaagcactga tagctgcccc gctgcgtatc ttcaacgcct 60
gtcggcttga gaaagacctg agtatgttgt aactaaaggt gcgcataatg tatattatgt 120
taaatggatc ctgggtatta aaaagaagat ctatttattt agagatctgt tctattgtga 180
tctcttatta ggatcgcact gccctgtgga taacaaggat ccggctttta agatcaacaa 240
cctggaaagg atcattaact gtgaatgatc ggtgatcctg gaccgtataa gctgggatca 300
gaatgagggg ttatacacaa ctcaaaaact gaacaacagt tgttctttgg ataactaccg 360
gttgatccaa gcttcctgac agagttatcc acagtagatc gcacgatctg tataacttcg 420
tatagcatac attatacgaa gttatcttta gttacaacat actccattct tctgccggat 480
cttcttacgc cccgccctgc cactcatcgc agtactgttg taattcatta agcattctgc 540
cgacatggaa gccatcacag acggcatgat gaacctgaat cgccagcggc atcagcacct 600
tgtcgccttg cgtataatat ttgcccatgg tgaaaacggg ggcgaagaag ttgtccatat 660
tggccacgtt taaatcaaaa ctggtgaaac tcacccaggg attggctgag acgaaaaaca 720
tattctcaat aaacccttta gggaaatagg ccaggttttc accgtaacac gccacatctt 780
gcgaatatat gtgtagaaac tgccggaaat cgtcgtggta ttcactccag agcgatgaaa 840
acgtttcagt ttgctcatgg aaaacggtgt aacaagggtg aacactatcc catatcacca 900
gctcaccgtc tttcattgcc atacggaatt ccggatgagc attcatcagg cgggcaagaa 960
tgtgaataaa ggccggataa aacttgtgct tatttttctt tacggtcttt aaaaaggccg 1020
taatatccag ctgaacggtc tggttatagg tacattgagc aactgactga aatgcctcaa 1080
aatgttcttt acgatgccat tgggatatat caacggtggt atatccagtg atttttttct 1140
ccattttagc ttccttagct cctgaaaatc tcgataactc aaaaaatacg cccggtagtg 1200
atcttatttc attatggtga aagttggaac ctcttacgtt ctaaaaccgt gactgcggat 1260
atcccgattg tgggggatgc tcgccaggtc ctcgaaca 1298
<210> 52
<211> 1509
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Km-oriC (DCW25)
<400> 52
gtctgcgtgg aaaaggtata aaagcactga tagctgcccc gctgcgtatc ttcaacgcct 60
gtcggcttga gaaagacctg agtatgttgt aactaaaggt gcgcataatg tatattatgt 120
taaatggatc ctgggtatta aaaagaagat ctatttattt agagatctgt tctattgtga 180
tctcttatta ggatcgcact gccctgtgga taacaaggat ccggctttta agatcaacaa 240
cctggaaagg atcattaact gtgaatgatc ggtgatcctg gaccgtataa gctgggatca 300
gaatgagggg ttatacacaa ctcaaaaact gaacaacagt tgttctttgg ataactaccg 360
gttgatccaa gcttcctgac agagttatcc acagtagatc gcacgatctg tataacttcg 420
tatagcatac attatacgaa gttatcttta gttacaacat actccattct tctgccggat 480
cttcgggaaa gccacgttgt gtctcaaaat ctctgatgtt acattgcaca agataaaaat 540
atatcatcat gaacaataaa actgtctgct tacataaaca gtaatacaag gggtgttatg 600
agccatattc aacgggaaac gtcttgctcg aggccgcgat taaattccaa catggatgct 660
gatttatatg ggtataaatg ggctcgcgat aatgtcgggc aatcaggtgc gacaatctat 720
cgattgtatg ggaagcccga tgcgccagag ttgtttctga aacatggcaa aggtagcgtt 780
gccaatgatg ttacagatga gatggtcaga ctaaactggc tgacggaatt tatgcctctt 840
ccgaccatca agcattttat ccgtactcct gatgatgcat ggttactcac cactgcgatc 900
cccgggaaaa cagcattcca ggtattagaa gaatatcctg attcaggtga aaatattgtt 960
gatgcgctgg cagtgttcct gcgccggttg cattcgattc ctgtttgtaa ttgtcctttt 1020
aacagcgatc gcgtatttcg tctcgctcag gcgcaatcac gaatgaataa cggtttggtt 1080
gatgcgagtg attttgatga cgagcgtaat ggctggcctg ttgaacaagt ctggaaagaa 1140
atgcataagc ttttgccatt ctcaccggat tcagtcgtca ctcatggtga tttctcactt 1200
gataacctta tttttgacga ggggaaatta ataggttgta ttgatgttgg acgagtcgga 1260
atcgcagacc gataccagga tcttgccatc ctatggaact gcctcggtga gttttctcct 1320
tcattacaga aacggctttt tcaaaaatat ggtattgata atcctgatat gaataaattg 1380
cagtttcatt tgatgctcga tgagtttttc taatcagaat tggttaattg gttgtaacac 1440
tggcagagct tctaaaaccg tgactgcgga tatcccgatt gtgggggatg ctcgccaggt 1500
cctcgaaca 1509
<210> 53
<211> 1509
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Km-oriC (DCW35)
<400> 53
agtcacatct taacggtgaa gctgaagtag aaaaacgtgt tacagcatca gttggctcgt 60
gtcggcttga gaaagacctg agtatgttgt aactaaaggt gcgcataatg tatattatgt 120
taaatggatc ctgggtatta aaaagaagat ctatttattt agagatctgt tctattgtga 180
tctcttatta ggatcgcact gccctgtgga taacaaggat ccggctttta agatcaacaa 240
cctggaaagg atcattaact gtgaatgatc ggtgatcctg gaccgtataa gctgggatca 300
gaatgagggg ttatacacaa ctcaaaaact gaacaacagt tgttctttgg ataactaccg 360
gttgatccaa gcttcctgac agagttatcc acagtagatc gcacgatctg tataacttcg 420
tatagcatac attatacgaa gttatcttta gttacaacat actccattct tctgccggat 480
cttcgggaaa gccacgttgt gtctcaaaat ctctgatgtt acattgcaca agataaaaat 540
atatcatcat gaacaataaa actgtctgct tacataaaca gtaatacaag gggtgttatg 600
agccatattc aacgggaaac gtcttgctcg aggccgcgat taaattccaa catggatgct 660
gatttatatg ggtataaatg ggctcgcgat aatgtcgggc aatcaggtgc gacaatctat 720
cgattgtatg ggaagcccga tgcgccagag ttgtttctga aacatggcaa aggtagcgtt 780
gccaatgatg ttacagatga gatggtcaga ctaaactggc tgacggaatt tatgcctctt 840
ccgaccatca agcattttat ccgtactcct gatgatgcat ggttactcac cactgcgatc 900
cccgggaaaa cagcattcca ggtattagaa gaatatcctg attcaggtga aaatattgtt 960
gatgcgctgg cagtgttcct gcgccggttg cattcgattc ctgtttgtaa ttgtcctttt 1020
aacagcgatc gcgtatttcg tctcgctcag gcgcaatcac gaatgaataa cggtttggtt 1080
gatgcgagtg attttgatga cgagcgtaat ggctggcctg ttgaacaagt ctggaaagaa 1140
atgcataagc ttttgccatt ctcaccggat tcagtcgtca ctcatggtga tttctcactt 1200
gataacctta tttttgacga ggggaaatta ataggttgta ttgatgttgg acgagtcgga 1260
atcgcagacc gataccagga tcttgccatc ctatggaact gcctcggtga gttttctcct 1320
tcattacaga aacggctttt tcaaaaatat ggtattgata atcctgatat gaataaattg 1380
cagtttcatt tgatgctcga tgagtttttc taatcagaat tggttaattg gttgtaacac 1440
tggcagagct tctaaaaccg tgactgcgga tatcccgatt gtgggggatg ctcgccaggt 1500
cctcgaaca 1509
<210> 54
<211> 3100
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Lter1
<400> 54
aatagaaaac tgccagtgcg caacctgtac cggttcatgc aatttaaaca gacaaatcgg 60
tctgccattg atcatatttt ctgacaaaag ctcgccacac tgttcaaacc cgcgacgcca 120
gcgttcagca gtggcgtttt gatggcaacg caaagaaatg tgatcggcag tcagcggagt 180
gatattcaac cccagacggc gggaaagttc atctaatgcg tggataaatc gcggtaaatc 240
cgatgcaata tcctgcagct cgtcgataga ttgccagttc gccataatca ctcttcgtct 300
ttcagtaaaa gcgttaattt accctgttgc cctgtgccaa ccaaccgctg atttcacgcc 360
gcttctgatg caatagtgaa aacggcaata cgccacgcgc acgttgctga cgaaaacagc 420
catttgcagt atactcccgc cctaatttct ttaactggtg cgggcaattt ttgctcgctt 480
catcaatgta aggtattccg gtgaatattc aggctcttct ctcagaaaaa gtccgtcagg 540
ccatgattgc ggcaggcgcg cctgcggatt gcgaaccgca ggttcgtcag tcagcaaaag 600
ttcagttcgg cgactatcag gctaacggca tgatggcagt tgctaaaaaa ctgggtatgg 660
caccgcgaca attagcagag caggtgctga ctcatctgga tcttaacggt atcgccagca 720
aagttgagat cgccggtcca ggctttatca acattttcct tgatccggca ttcctggctg 780
aacatgttca gcaggcgctg gcgtccgatc gtctcggtgt tgctacgcca gaaaaacaga 840
ccattgtggt tgactactct gcgccaaacg tggcgaaaga gatgcatgtc ggtcacctgc 900
gctctaccat tattggtgac gcagcagtgc gtactctgga gttcctcggt cacaaagtga 960
ttcgcgcaaa ccacgtcggc gactggggca ctcagttcgg tatgctgatt gcatggctgg 1020
aaaagcagca gcaggaaaac gccggtgaaa tggagctggc tgaccttgaa ggtttctacc 1080
gcgatgcgaa aaagcattac gatgaagatg aagagttcgc cgagcgcgca cgtaactacg 1140
tggtaaaact gcaaagcggt gacgaatatt tccgcgagat gtggcgcaaa ctggtcgaca 1200
tcaccatgac gcagaaccag atcacctacg atcgtctcaa cgtgacgctg acccgtgatg 1260
acgtgatggg cgaaagcctc tacaacccga tgctgccagg aattgtggcg gatctcaaag 1320
ccaaaggtct ggcagtagaa agcgaagggg cgaccgtcgt attccttgat gagtttaaaa 1380
acaaggaagg cgaaccgatg ggcgtgatca ttcagaagaa agatggcggc tatctctaca 1440
ccaccactga tatcgcctgt gcgaaatatc gttatgaaac actgcatgcc gatcgcgtgc 1500
tgtattacat cgactcccgt cagcatcaac acctgatgca ggcatgggcg atcgtccgta 1560
aagcaggcta tgtaccggaa tccgtaccgc tggaacacca catgttcggc atgatgctgg 1620
gtaaagacgg caaaccgttc aaaacccgcg cgggtggtac agtgaaactg gccgatctgc 1680
tggatgaagc cctggaacgt gcacgccgtc tggtggcaga aaagaacccg gatatgccag 1740
ccgacgagct ggaaaaactg gctaacgcgg ttggtattgg tgcggtgaaa tatgcggatc 1800
tctccaaaaa ccgcaccacg gactacatct tcgactggga caacatgctg gcgtttgagg 1860
gtaataccgc gccatacatg cagtatgcat acacgcgtgt attgtccgtg ttccgtaaag 1920
cagaaattga cgaagagcaa ctggctgcag ctccggttat catccgtgaa gatcgtgaag 1980
cgcaactggc agctcgcctg ctgcagtttg aagaaaccct caccgtggtt gcccgtgaag 2040
gcacgccgca tgtaatgtgt gcttacctgt acgatctggc cggtctgttc tctggcttct 2100
acgagcactg cccgatcctc agcgcagaaa acgaagaagt gcgtaacagc cgtctaaaac 2160
tggcacaact gacggcgaag acgctgaagc tgggtctgga tacgctgggt attgagactg 2220
tagagcgtat gtaatcgatt tttcgtgaga gtgaagcctg atcatgacct ggcaaagcag 2280
aaagcacaat cctcaggcgg gttaccgggt caaaggtggt cgtccggcgc tggtggtggt 2340
gtttctctgt ggtattgctg tgattggcgt gcaatttttg attgcggcag ggttgttacc 2400
agaagtgggg tgatcagata gcctcaaatt ccttattggg tgccagaatt aacgctgaca 2460
cccaatttgg cctcttaatg caggcagcac tgcttaaatt tcttaccact accgcacggg 2520
caaggatcgt tacgccccgg tttctcttct gctttgatcg gttgctgaac agctttttcc 2580
tgcggatgcg ccatccagta cgcatgtaga tcaagcgccg ccagtcgaat ggcatctacg 2640
ctctcttcaa acgcttctgg cgacatcttt tctacccgct cgaagttttc ctcagtaccg 2700
tgcagcgcaa tcgcctccag cgctggtttt aacgaatcgg gcaacgttga ccagtcagaa 2760
agtgccacgc cccgcatata gccaaagcac cactcctcaa caatcgtcag ctcgctgcca 2820
tcaacttctc gcaagccgaa taacggctca aactgctccg ggaattcgtt cagacgctct 2880
gcggtatcgg ccatatgttg aaaagccaga ttcataaagc gcgtcatctc tttctctgac 2940
gcccagcgcg gcacatagtc agccccaccc cacacggcaa ccagccactg ttccggttca 3000
atctcttgcg gagaactcaa caccgccgtc aataaaccgt ccagctccgc cacatcaagg 3060
atggcgtggt cagtgttgta tttggtcaga atatcgtcca 3100
<210> 55
<211> 3100
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Lter2
<400> 55
ccagctccgc cacatcaagg atggcgtggt cagtgttgta tttggtcaga atatcgtcca 60
gccattccaa ctcactttcg tttaacggtc ccgttttcat acgcttttcc ttgtggatct 120
caactcgcca gcacctatct tacatgccgg tccgtatcag agatactttt tgagtggctt 180
tgctggtgat taaaaattaa ggagggtgta acgacaagtt gcaggcacaa aaaaaccacc 240
cgaaggtggt ttcacgacac tgcttattgc tttgatttta ttcttatctt tcccatggta 300
cccggagcgg gacttgaacc cgcacagcgc gaacgccgag ggattttaaa tcccttgtgt 360
ctaccgattc caccatccgg gctcgggaag aaagtggagg cgcgttccgg agtcgaaccg 420
gactagacgg atttgcaatc cgctacataa ccgctttgtt aacgcgccaa attcttcagg 480
cctttcagcc agacatccgc ttgacgccga tgtcttttaa actggagcgg gaaacgagac 540
tcgaactcgc gaccccgacc ttggcaaggt cgtgctctac caactgagct attcccgcat 600
tcatcaagca atcagttaat cacttgattt tattatcgtc tggcaatcag tgccgccgtt 660
cgatgcgttg cattctactt acctggcgcg atgagtcaac gatatttttc accacttttg 720
atcgtttgct gaaaattacg ccgaaacgat cactgatcaa gcaaatctgc acgcgcagcg 780
ctcaaatatt gcaacattga ccacagagtc agtaccgcag ccacaaagaa aagtgcaata 840
ccggcgtact caacccaaat gttcggacgc cacagcagcc atgccaacgc caccatctgg 900
gcagtggttt tcactttccc aatccaggag acggccacgc tactgcgttt acccaactcc 960
gccatccatt cgcgtagcgc agaaataata atttcacggg cgatcatcgt tgccgccggt 1020
aatgtcaccc accagctgtg ataatgctcg gttaccagca ccatggcgat agccacgaga 1080
actttatctg ccacagggtc aaggaaagca ccaaaccggg tactctggtt ccagcggcgt 1140
gccagaaaac catcgaacca gtcagtcacc gccgcgacgc agaaaatgag cgcggcggca 1200
aacggcgacc aggtgacagg cagataaaag accaatacaa agaatgggat aaggatgaca 1260
cggaacagtg taagcaacgt agggatatta aattgcataa tgacgggtaa ctatctgttg 1320
tcagtaagat tacccctatg ttgctacaga gacatcaatg tttcaacgac cagaagatct 1380
tttctgccag accttgcgaa atacccggca cttttaatcg tgggctgttt gcttccttgg 1440
gcggatacga gttttattat cgtcttaatg atttccacat attaaaagca agtatgcttt 1500
caaaacacaa ttataaaaaa tcccgccaac aatataagtt tttataaaat taaatataag 1560
attatggctt tagaatattt ttatttctaa tagacgagat ttttcctgtt atgatataat 1620
atgctgaatt aacacatgtt aacgatttac cagtaatgta aataaatttt cgaggagatc 1680
attccagtgg gacgtaaatg ggccaatatt gttgctaaaa aaacggctaa agacggtgca 1740
acgtctaaaa tttatgcaaa attcggtgta gaaatctatg ctgctgctaa acaaggtgaa 1800
cccgatccag aattaaacac atctttaaaa ttcgttattg aacgtgcaaa gcaggcacaa 1860
gttccaaagc acgttattga taaagcaatt gataaagcca aaggcggcgg agatgaaacg 1920
ttcgtgcagg gacgttatga aggctttggt cctaatggct caatgattat cgccgagaca 1980
ttgacttcaa atgttaaccg tacgattgct aacgttcgca caattttcaa taaaaaaggc 2040
ggcaatatcg gagcggcagg ttctgtcagc tatatgtttg acaatacggg tgtgattgta 2100
tttaaaggga cagaccctga ccatattttt gaaattttac ttgaagctga agttgatgtt 2160
cgtgatgtga ctgaagaaga aggtaacatt gttatttata ctgaacctac tgaccttcat 2220
aaaggaatcg cggctctaaa agcagctgga atcactgagt tctcaacaac agaattagaa 2280
atgattgctc aatctgaagt tgagctttcc ccagaagatt tagaaatctt tgaagggctt 2340
gttgatgccc ttgaagatga cgacgatgta caaaaagttt atcataacgt cgcaaatctc 2400
taattatctt ttaaagaaat ctgtctttac ggcagatttc tttaatctca tataattctt 2460
ataaaaaata taatattcaa ctcgtcatat tgattatacc cccccgttcc cagagaaata 2520
atatttatta aaattccagt tcttcttttt ctgattacag aaggcaaagt ggcaattacg 2580
catagtttcc cgataaagac gcgatagcga catcccgcat aaggcatttt tctctttatc 2640
tttgtacggt acttcatgga acagagtttt tgaccttgcg aatcgtgatg tctgttgggg 2700
agggacaatt tgctcactga agcgtgagac tcgattaagc gcacgaaaca cagaaatcaa 2760
aaaacccggt cactttttta caaggtaacc gggtaaaaat aatttttatt ttttaactgt 2820
tttgagactc atagagatgt ctcaaaacta aaatttggct cctctgactg gactcgaacc 2880
agtgacatac ggattaacag tccgccgttc taccgactga actacagagg aatcgtgaga 2940
acgaggcgaa tattagcgat gcccacccac aatgtcaaag cctgtttttt aaatttgaaa 3000
tcgtttgctg aaataatctg cattttgtcg tttattccga cacaactggc tttttttcac 3060
acttttgcgg ctcgggtcga gggtatttcc atagccaacg 3100
<210> 56
<211> 3100
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Lter3
<400> 56
cacaactggc tttttttcac acttttgcgg ctcgggtcga gggtatttcc atagccaacg 60
tccagtaacc attcgccagt aaaacagcac ggcccgcaca gcccagtcgg caaacattcc 120
catccagaca ccaaccacac cccagccaag catgattccc agcacataac cgactacaac 180
ccgacaaccc cacatgctca acatcgaaac ccacatggcg taacgggcat cacgagcacc 240
tttaaatcca gcgggtagca cccatgaggc ggaccaaata ggcataaata aagcatttag 300
ccaaatcaga atcacaacga catgtttaac ctgtggatcc tgggtgtaaa acgatgccat 360
aaccccggca aagggagccg ttagccaggc gatggccgtt aatccaagag tggaaagcca 420
gaacacatgc cgcaactgaa tctctgcttg cgctatctgc cctaccccca accttcggcc 480
tgtaatgatc gtagaagcag agccgagcgc acttccgggt aagttgataa gagccgcaat 540
tgaaaacgcg ataaaatttc cggcaataac actggtcccc atcccggcaa cgaacatttg 600
ggttaataac cgaccactgg taaataacac tgattcgaca ctcgcgggaa taccaatccc 660
catgacttcc cagataatgc taaaattcag cggtttaaaa tagctcttta acgaaatcct 720
tagcgcagga ttaaaaccaa tcgccagcac ccacaaaatt gcaactgcgc caatataacg 780
agaaatggtt aaacccagcc ctgccccgac aaatcccagt cccggccagg agaaaaggcc 840
gtaaatcaat atgccgctaa taataatatt aagaatattc aggctaccgt taatcaatag 900
cggtattttc gtattccctg caccacgaag tgccccgcta ccaataagag tgatggcagc 960
tgctggataa ctgagtaccg tcagctccag ataagtcaac gccagtgctt taacttctgt 1020
cgtggcatca cccgcgacga aatcaataat ttgttcgcca aaatgatgaa taagcgttgc 1080
caacagtacg gcaaacaacg tcatgatcac caatgactgc cgcgtcgcca ccctcgctcg 1140
tcgtcgatcc cgcttaccga gactaaatgc cacaacgaca gtagtaccaa gatcgatagc 1200
agcaaaaaaa gccataatga ccatattgaa gctgtccgcc aatcccacgc cggccatcgc 1260
atcttttccc agccagctga ccagaaaagt gctcagaacc cccatcaaca ggacacaggc 1320
attctccatg aagataggaa cagcaagcgg ggttatctcg cgccagaaca acactttgta 1380
gctcttgcgt ttagcgtgcc agcgagtgcc gtgaacaacc tggcgtaaag cagaggagat 1440
attcaaagcc gaccttaatt gcagaaagtg aaaccacatt tcaaataatg agggagaatc 1500
agcaaagctg caaagatttt cgccaacaaa ttgtctgcaa atgcaacaaa ctgttgatag 1560
aaacggcaaa cagttgggga atttaaaaat cgggtttgac aaaagatttt tcgccgttaa 1620
gatgtgcctc aacaacgatt cctctgtagt tcagtcggta gaacggcgga ctgttaatcc 1680
gtatgtcact ggttcgagtc cagtcagagg agccaaattc ctgaaaagcc cgcttttata 1740
gcgggatttt tgctatatct gataatcaat ttcctcttca ctgctttcca tcacctgccg 1800
cttgatatcc tcaactgaca gtcctgcatt acaaagttcc agaaagcgcc agacatagtt 1860
acgctgaagt tgtcctcgct tcagtcccaa ccagacagta ttagcatcaa aaagatgccg 1920
cgtatccagg cggattaaat tctcttcctc ttgttcgcca ctggattgct cggcaactaa 1980
tccgatccca agcccaagag caacataggt tttaatgaca tcagaatcct gcgcacttaa 2040
tacaatatct gccagcaaac ctttgcgggc aaatgcgtca tcaatacgtg agcgccccgt 2100
aatcccctgt cggtaagtga ttaacggcca cttcgctatt gattccagcg tcaatggtga 2160
aatttgcgtc aagggatgat cgtgtggaac aagcaaacta tggtgccaac gaaaccacgg 2220
gaaggcgacg agctgcgggt cattactcaa acgctcgctg gcgataccaa tatcagcttc 2280
gccattttgc aacaatgtcg caatttcctg tggcgtcccc tggattagct cgagccgaac 2340
ctccgggaaa agttcgcgaa aagctttaat gacctctggc aagctataac gtgcctgagt 2400
atgcgtcgtt gcaatagtga gaacgccaga cgtatcgttg gtaaacaggt ctgcaagccg 2460
acgaacatta ctggcttcat tcagaatacg ttctgcaatg accagtaatg ctttgcccgg 2520
ttcagtcatg cccagcagtc gcttacctcg tcgaacaaat atttcgatgc caagttcatc 2580
ctccagttcc cgaatatgac ggctgacgcc tgactgtgag gtaaaaagca tattcgcaac 2640
ctctgtcagg ttgtaatcct gacgtgcagc ctcgcggatt atctttagtt gttggaaatt 2700
cacggtaaac tccgggcagt tcagatttcc cgttattgtt aaagtctaat gcccggcata 2760
acaaataata aaaacccgca tcttattcca tcccgatata acacttagct caccaattgc 2820
cactgccttt tttccatcac tggagaacta atcactgaca ttaacaactc tttcactgcc 2880
tgtgcctgtg gcgataagtt cgctctggcg ggtaaattta atgacaaaga gagactcatg 2940
gaaggagtgg taatgcgtga catccaccca tttactgcgc cacataacga acgcgcggcc 3000
gattcgggta atactgcaac gcccatgccg ctggcaatcg ctgcggtaag cgtggcaata 3060
gactcaattt caccaataac ttttgccgtg agtcgccgta 3100
<210> 57
<211> 3100
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Lter4
<400> 57
ctgcggtaag cgtggcaata gactcaattt caccaataac ttttgccgtg agtcgccgta 60
gggaaaaagc ctcatcaaca cgaagtctaa tagcactgta atcactgggg agaaagaggt 120
tcatttgcgc aatagcattc acatcaacgc tttgccccgg gcaatcttga gttcctacca 180
gaaaaagatc ttctttcagc aaagcctgac tggatacacc agccacaggg gaatgctcat 240
aaatcaccgc catatcgagt tggtgattta tcaatttttc gttaagcact gcaccactat 300
tttcatgaag atagataacg atctccggaa attcagcgcg aaccgcctgt aataagggca 360
tggtgatgga tgacgcagcg gttcctggtg caaagccaat cgagacttgc cccgataatg 420
cctgaccaac gttatgcacc gccagttggg cctgttcaca ctgacgtaaa atggcccgcg 480
catgggtata gagaattttt ccggcgtctg ttggtgtaac gccccgcttt gtacggatca 540
aaagttgttg atttaactca ccttccagtg tggcaacctg ctggctgagc gctggttgtg 600
cgatatgcaa tacttcagca gcctgggtca ggctaccaat atctacaatt tttacgaagt 660
atttcaggcg tctgaagttc atgttgcctc cggtttttaa gaatcggccc aagtgccgcc 720
attacttaca accagattgc aagatgcttg ccagttttat tttggtgttg atgtacaagc 780
taaccaactg tcaaataaga gattatgata gattcgtcat ttgctccttt aatcagctgt 840
cgcgttcccc tgccctataa aaggagggta tgcaccacga tggttcatta cccaataaga 900
ttgaaagctc accactttgt tgaaattgac agcaaacaaa caaaaaaatg catttcaccc 960
tttgacatca ccatgcactg ccattaatat gcgccccgtt cacacgattc ctctgtagtt 1020
cagtcggtag aacggcggac tgttaatccg tatgtcactg gttcgagtcc agtcagagga 1080
gccaaattca aaaaagcctg ctttctagca ggctttttgc tttctaatta ccaacgctct 1140
taaaacatct gtcttgaacc agaactaatt tgcacaggca ttcccgatcg acgttgcaac 1200
gcagcatttg cgcgatttac atcaacttct tgcccgttga taaacgcccg caaagatggg 1260
gttaccggca atggcacttt tcggtcagac tcatattctg cacgattgcg cgacaatggc 1320
tcatgaactt ccagccagtt cgagccatct ggttcagtgg tgtattttac tggctggtcg 1380
ataatttgca cacgcgtccc aacaggaaca ttatcaaaca gatatttgat atcgtcattg 1440
cgcagacgaa tacagccctg acttacccgg agcccaatac caaaattggc attggtacca 1500
tggatggcat acaacctgcc aatataaatc gcgtacagcc ccatgggatt atcggggccc 1560
gcaggaacaa atgcgggcaa actctcccct cgtttcgcat attcgcgccg agtgttcggc 1620
gttggcgtcc aggttggagc ttcttgttta cgttcaacgg tagtcaccca gttacgcggg 1680
gtttctcgcc cagcctggcc gataccaata ggaaagactt ccacagtatt actgtctggt 1740
gggtagtaat aaagacgcat ctcagcgacg ttaacaacaa tccctttacg aacagtgtcg 1800
ggcaaaatca gttgctgcgg aatggtgagt tgcgagccag acttcggcaa aaaaacatca 1860
gcgcccgggt tcgcttccag catgttactt aacccttgcc cgtattgtgc ggcaaaagtc 1920
tccagcggct gggtattgtg atcaggaaca gttacagtaa acgactgccc cactaaacgg 1980
ctaccctctg gaggtaatgg ataagttacc gccaggctag tatggctggc aaaaagcaga 2040
gcaaatgagc aaagaatatt tacacgacgc atcatgtccc tttcctatgt cgcgaaagct 2100
atccgttaag tatagctttt atcagacttt tcgtttttaa ctgttcaaat cagaagtcgt 2160
attccccggt agaacattgt tcgttcatcc cgactccttt tttgtataga taaaccatca 2220
gctgatagtt tacctgaaga atatagagaa gtacttactt aacattttcc catttggtac 2280
tatctaaccc cttttcacta ttaagaagta atgcctacta tgactcaagt cgcgaagaaa 2340
attctggtga cgtgcgcact gccgtacgct aacggctcaa tccacctcgg ccatatgctg 2400
gagcacatcc aggctgatgt ctgggtccgt taccagcgaa tgcgcggcca cgaggtcaac 2460
ttcatctgcg ccgacgatgc ccacggtaca ccgatcatgc tgaaagctca gcagcttggt 2520
atcaccccgg agcagatgat tggcgaaatg agtcaggagc atcagactga tttcgcaggc 2580
tttaacatca gctatgacaa ctatcactcg acgcacagcg aagagaaccg ccagttgtca 2640
gaacttatct actctcgcct gaaagaaaac ggttttatta aaaaccgcac catctctcag 2700
ctgtacgatc cggaaaaagg catgttcctg ccggaccgtt ttgtgaaagg cacctgcccg 2760
aaatgtaaat ccccggatca atacggcgat aactgcgaag tctgcggcgc gacctacagc 2820
ccgactgaac tgatcgagcc gaaatcggtg gtttctggcg ctacgccggt aatgcgtgat 2880
tctgaacact tcttctttga tctgccctct ttcagcgaaa tgttgcaggc atggacccgc 2940
agcggtgcgt tgcaggagca ggtggcaaat aaaatgcagg agtggtttga atctggcctg 3000
caacagtggg atatctcccg cgacgcccct tacttcggtt ttgaaattcc gaacgcgccg 3060
ggcaaatatt tctacgtctg gctggacgca ccgattggct 3100
<210> 58
<211> 2650
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Lter5
<400> 58
ttgaaattcc gaacgcgccg ggcaaatatt tctacgtctg gctggacgca ccgattggct 60
acatgggttc tttcaagaat ctgtgcgaca agcgcggcga cagcgtaagc ttcgatgaat 120
actggaagaa agactccacc gccgagctgt accacttcat cggtaaagat attgtttact 180
tccacagcct gttctggcct gccatgctgg aaggcagcaa cttccgcaag ccgtccaacc 240
tgtttgttca tggctatgtg acggtgaacg gcgcaaagat gtccaagtct cgcggcacct 300
ttattaaagc cagcacctgg ctgaatcatt ttgacgcaga cagcctgcgt tactactaca 360
ctgcgaaact ctcttcgcgc attgatgata tcgatctcaa cctggaagat ttcgttcagc 420
gtgtgaatgc cgatatcgtt aacaaagtgg ttaacctggc ctcccgtaat gcgggcttta 480
tcaacaagcg ttttgacggc gtgctggcaa gcgaactggc tgacccgcag ttgtacaaaa 540
ccttcactga tgccgctgaa gtgattggtg aagcgtggga aagccgtgaa tttggtaaag 600
ccgtgcgcga aatcatggcg ctggctgatc tggctaaccg ctatgtcgat gaacaggctc 660
cgtgggtggt ggcgaaacag gaaggccgcg atgccgacct gcaggcaatt tgctcaatgg 720
gcatcaacct gttccgcgtg ctgatgactt acctgaagcc ggtactgccg aaactgaccg 780
agcgtgcaga agcattcctc aatacggaac tgacctggga tggtatccag caaccgctgc 840
tgggccacaa agtgaatccg ttcaaggcgc tgtataaccg catcgatatg aggcaggttg 900
aagcactggt ggaagcctct aaagaagaag taaaagccgc tgccgcgccg gtaactggcc 960
cgctggcaga tgatccgatt caggaaacca tcacctttga cgacttcgct aaagttgacc 1020
tgcgcgtggc gctgattgaa aacgcagagt ttgttgaagg ttctgacaaa ctgctgcgcc 1080
tgacgctgga tctcggcggt gaaaaacgca atgtcttctc cggtattcgt tctgcttacc 1140
cggatccgca ggcactgatt ggtcgtcaca ccattatggt ggctaacctg gcaccacgta 1200
aaatgcgctt cggtatctct gaaggcatgg tgatggctgc cggtcctggc gggaaagata 1260
ttttcctgct aagcccggat gccggtgcta aaccgggtca tcaggtgaaa taatccccct 1320
tcaaggcgct gcatcgacag cgccttttct ttataaattc ctaaagttgt tttcttgcga 1380
ttttgtctct ctctaacccg cataaaatcg agcgtgacgt tgcgctccat ggttcctgcc 1440
ttttaatcag ttgtgatgac gcacagcgcg cagaaactcg tggcgcgtat tctgactgga 1500
tttgaacaat ccaccaagag aggtcgttgt cgtggcactg gttgcatcgc ggatgccacg 1560
cgccttcacg cagtaatgca ccgcgtcgat cgagacagcc acgttattgg tgcccagcag 1620
cgtttgtagc gcaataagaa tttgctgcgt cagacgttcc tgcacctgcg gacgctgggc 1680
aaagaactgc acaatgcggt taatttttga cagaccgatc accgaatctt tcgggatata 1740
ggccaccgtc gctttgccat cgatggtaac aaaatggtgt tcacaggtgc tggtcagagt 1800
gatatcgcgc acggtgacca tttcatcgac cttcattttg ttttcaatga gggtgatttt 1860
cgggaaattg gcgtaatcca gaccggagaa aatttcatcg acatacattt tagcgatgcg 1920
atgcggcgtt tccatcaaac tgtcatcagc caggtcgaga ttcagcagct gcatgatttc 1980
ggtcatatga ccagcaataa ggcttttgcg cgtttcgtta tccatttcat gcacgggcgg 2040
gcgcagcggt gtttccagtc ctcgcgcaac taacgcttca tgaaccaggg ccgcttcttt 2100
actgagtgat ggcatttatg atttctcctg caggtgtgac gcctccgccc tgcgtggggg 2160
caaagttatt aagctgattt acagcctgat tattgtgcgt gaggcggcgc acataatcca 2220
gtattcacag cgataattat tgtaattgcc gctgcctttc atcagcagat gttaaaacat 2280
cgttatgcaa atacggaagt gaaagttact cacagcacat tgaataaacg gtatgatgaa 2340
gaaattgcaa acaacacaac aaggagccac gcatggaaat gctcgaagag caccgctgtt 2400
ttgaaggctg gcagcaacgc tggcgacacg actccagtac cttaaactgc ccgatgacgt 2460
tcagtatctt tctccctcca cctcgtgatc acactccgcc accagtgctg tactggcttt 2520
ccggattaac ctgcaatgac gagaacttca ccaccaaggc gggtgcccag cgggtagcgg 2580
cggaactggg gattgtactg gtgatgccag acaccagccc gcgcggcgaa aaggttgcca 2640
acgacgatgg 2650
<210> 59
<211> 1298
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Фрагмент Cm-oriC/325k
<400> 59
tattggtaac cagaccggca ttttacgtaa tgaaccaggc atcctttctc ccacaaatat 60
cgtaagaggt tccaactttc accataatga aataagatca ctaccgggcg tattttttga 120
gttatcgaga ttttcaggag ctaaggaagc taaaatggag aaaaaaatca ctggatatac 180
caccgttgat atatcccaat ggcatcgtaa agaacatttt gaggcatttc agtcagttgc 240
tcaatgtacc tataaccaga ccgttcagct ggatattacg gcctttttaa agaccgtaaa 300
gaaaaataag cacaagtttt atccggcctt tattcacatt cttgcccgcc tgatgaatgc 360
tcatccggaa ttccgtatgg caatgaaaga cggtgagctg gtgatatggg atagtgttca 420
cccttgttac accgttttcc atgagcaaac tgaaacgttt tcatcgctct ggagtgaata 480
ccacgacgat ttccggcagt ttctacacat atattcgcaa gatgtggcgt gttacggtga 540
aaacctggcc tatttcccta aagggtttat tgagaatatg tttttcgtct cagccaatcc 600
ctgggtgagt ttcaccagtt ttgatttaaa cgtggccaat atggacaact tcttcgcccc 660
cgttttcacc atgggcaaat attatacgca aggcgacaag gtgctgatgc cgctggcgat 720
tcaggttcat catgccgtct gtgatggctt ccatgtcggc agaatgctta atgaattaca 780
acagtactgc gatgagtggc agggcggggc gtaagaagat ccggcagaag aatggagtat 840
gttgtaacta aagataactt cgtataatgt atgctatacg aagttataca gatcgtgcga 900
tctactgtgg ataactctgt caggaagctt ggatcaaccg gtagttatcc aaagaacaac 960
tgttgttcag tttttgagtt gtgtataacc cctcattctg atcccagctt atacggtcca 1020
ggatcaccga tcattcacag ttaatgatcc tttccaggtt gttgatctta aaagccggat 1080
ccttgttatc cacagggcag tgcgatccta ataagagatc acaatagaac agatctctaa 1140
ataaatagat cttcttttta atacccagga tccatttaac ataatataca ttatgcgcac 1200
ctttagttac aacatactca ggtctttctc aagccgacct agagaataaa tttatattga 1260
ttaaatgaat gtatatttca aattgatttt gtttgtta 1298
<210> 60
<211> 1298
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Фрагмент Cm-oriC/220k
<400> 60
caaaactgac ataaatctcc agagatgtgt tcaggagtta gaaagattat ttcttctatt 60
cgtaagaggt tccaactttc accataatga aataagatca ctaccgggcg tattttttga 120
gttatcgaga ttttcaggag ctaaggaagc taaaatggag aaaaaaatca ctggatatac 180
caccgttgat atatcccaat ggcatcgtaa agaacatttt gaggcatttc agtcagttgc 240
tcaatgtacc tataaccaga ccgttcagct ggatattacg gcctttttaa agaccgtaaa 300
gaaaaataag cacaagtttt atccggcctt tattcacatt cttgcccgcc tgatgaatgc 360
tcatccggaa ttccgtatgg caatgaaaga cggtgagctg gtgatatggg atagtgttca 420
cccttgttac accgttttcc atgagcaaac tgaaacgttt tcatcgctct ggagtgaata 480
ccacgacgat ttccggcagt ttctacacat atattcgcaa gatgtggcgt gttacggtga 540
aaacctggcc tatttcccta aagggtttat tgagaatatg tttttcgtct cagccaatcc 600
ctgggtgagt ttcaccagtt ttgatttaaa cgtggccaat atggacaact tcttcgcccc 660
cgttttcacc atgggcaaat attatacgca aggcgacaag gtgctgatgc cgctggcgat 720
tcaggttcat catgccgtct gtgatggctt ccatgtcggc agaatgctta atgaattaca 780
acagtactgc gatgagtggc agggcggggc gtaagaagat ccggcagaag aatggagtat 840
gttgtaacta aagataactt cgtataatgt atgctatacg aagttataca gatcgtgcga 900
tctactgtgg ataactctgt caggaagctt ggatcaaccg gtagttatcc aaagaacaac 960
tgttgttcag tttttgagtt gtgtataacc cctcattctg atcccagctt atacggtcca 1020
ggatcaccga tcattcacag ttaatgatcc tttccaggtt gttgatctta aaagccggat 1080
ccttgttatc cacagggcag tgcgatccta ataagagatc acaatagaac agatctctaa 1140
ataaatagat cttcttttta atacccagga tccatttaac ataatataca ttatgcgcac 1200
ctttagttac aacatactca ggtctttctc aagccgacct agaagcgatc actgctgggt 1260
tacctgtttt aacaacagcg gtatgtgggt acgcgcat 1298
<210> 61
<211> 353
<212> PRT
<213> Escherichia coli
<220>
<223> RecA
<400> 61
Met Ala Ile Asp Glu Asn Lys Gln Lys Ala Leu Ala Ala Ala Leu Gly
1 5 10 15
Gln Ile Glu Lys Gln Phe Gly Lys Gly Ser Ile Met Arg Leu Gly Glu
20 25 30
Asp Arg Ser Met Asp Val Glu Thr Ile Ser Thr Gly Ser Leu Ser Leu
35 40 45
Asp Ile Ala Leu Gly Ala Gly Gly Leu Pro Met Gly Arg Ile Val Glu
50 55 60
Ile Tyr Gly Pro Glu Ser Ser Gly Lys Thr Thr Leu Thr Leu Gln Val
65 70 75 80
Ile Ala Ala Ala Gln Arg Glu Gly Lys Thr Cys Ala Phe Ile Asp Ala
85 90 95
Glu His Ala Leu Asp Pro Ile Tyr Ala Arg Lys Leu Gly Val Asp Ile
100 105 110
Asp Asn Leu Leu Cys Ser Gln Pro Asp Thr Gly Glu Gln Ala Leu Glu
115 120 125
Ile Cys Asp Ala Leu Ala Arg Ser Gly Ala Val Asp Val Ile Val Val
130 135 140
Asp Ser Val Ala Ala Leu Thr Pro Lys Ala Glu Ile Glu Gly Glu Ile
145 150 155 160
Gly Asp Ser His Met Gly Leu Ala Ala Arg Met Met Ser Gln Ala Met
165 170 175
Arg Lys Leu Ala Gly Asn Leu Lys Gln Ser Asn Thr Leu Leu Ile Phe
180 185 190
Ile Asn Gln Ile Arg Met Lys Ile Gly Val Met Phe Gly Asn Pro Glu
195 200 205
Thr Thr Thr Gly Gly Asn Ala Leu Lys Phe Tyr Ala Ser Val Arg Leu
210 215 220
Asp Ile Arg Arg Ile Gly Ala Val Lys Glu Gly Glu Asn Val Val Gly
225 230 235 240
Ser Glu Thr Arg Val Lys Val Val Lys Asn Lys Ile Ala Ala Pro Phe
245 250 255
Lys Gln Ala Glu Phe Gln Ile Leu Tyr Gly Glu Gly Ile Asn Phe Tyr
260 265 270
Gly Glu Leu Val Asp Leu Gly Val Lys Glu Lys Leu Ile Glu Lys Ala
275 280 285
Gly Ala Trp Tyr Ser Tyr Lys Gly Glu Lys Ile Gly Gln Gly Lys Ala
290 295 300
Asn Ala Thr Ala Trp Leu Lys Asp Asn Pro Glu Thr Ala Lys Glu Ile
305 310 315 320
Glu Lys Lys Val Arg Glu Leu Leu Leu Ser Asn Pro Asn Ser Thr Pro
325 330 335
Asp Phe Ser Val Asp Asp Ser Glu Gly Val Ala Glu Thr Asn Glu Asp
340 345 350
Phe
<210> 62
<211> 1509
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Km-oriC (DCW49)
<400> 62
agtcacatct taacggtgaa gctgaagtag aaaaacgtgt tacagcatca gttggctcgt 60
gtcggcttga gaaagacctg agtatgttgt aactaaaggt gcgcataatg tatattatgt 120
taaatggatc ctgggtatta aaaagaagat ctatttattt agagatctgt tctattgtga 180
tctcttatta ggatcgcact gccctgtgga taacaaggat ccggctttta agatcaacaa 240
cctggaaagg atcattaact gtgaatgatc ggtgatcctg gaccgtataa gctgggatca 300
gaatgagggg ttatacacaa ctcaaaaact gaacaacagt tgttctttgg ataactaccg 360
gttgatccaa gcttcctgac agagttatcc acagtagatc gcacgatctg tataacttcg 420
tatagcatac attatacgaa gttatcttta gttacaacat actccattct tctgccggat 480
cttcgggaaa gccacgttgt gtctcaaaat ctctgatgtt acattgcaca agataaaaat 540
atatcatcat gaacaataaa actgtctgct tacataaaca gtaatacaag gggtgttatg 600
agccatattc aacgggaaac gtcttgctcg aggccgcgat taaattccaa catggatgct 660
gatttatatg ggtataaatg ggctcgcgat aatgtcgggc aatcaggtgc gacaatctat 720
cgattgtatg ggaagcccga tgcgccagag ttgtttctga aacatggcaa aggtagcgtt 780
gccaatgatg ttacagatga gatggtcaga ctaaactggc tgacggaatt tatgcctctt 840
ccgaccatca agcattttat ccgtactcct gatgatgcat ggttactcac cactgcgatc 900
cccgggaaaa cagcattcca ggtattagaa gaatatcctg attcaggtga aaatattgtt 960
gatgcgctgg cagtgttcct gcgccggttg cattcgattc ctgtttgtaa ttgtcctttt 1020
aacagcgatc gcgtatttcg tctcgctcag gcgcaatcac gaatgaataa cggtttggtt 1080
gatgcgagtg attttgatga cgagcgtaat ggctggcctg ttgaacaagt ctggaaagaa 1140
atgcataagc ttttgccatt ctcaccggat tcagtcgtca ctcatggtga tttctcactt 1200
gataacctta tttttgacga ggggaaatta ataggttgta ttgatgttgg acgagtcgga 1260
atcgcagacc gataccagga tcttgccatc ctatggaact gcctcggtga gttttctcct 1320
tcattacaga aacggctttt tcaaaaatat ggtattgata atcctgatat gaataaattg 1380
cagtttcatt tgatgctcga tgagtttttc taatcagaat tggttaattg gttgtaacac 1440
tggcagagct tctaaaaccg tgactgcgga tatcccgatt gtgggggatg ctcgccaggt 1500
cctcgaaca 1509
<210> 63
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 63
ctatgcggca tcagagcag 19
<210> 64
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 64
gttaagccag ccccgacac 19
<210> 65
<211> 500
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 65
cgcgtcagcg ggtgttggcg ggtgtcgggg ctggcttaac agtactgcga tgagtggcag 60
ggcggggcgt aattttttta aggcagttat tggtgccctt aaacgcctgg ttgctacgcc 120
tgaataagtg ataataagcg gatgaatggc agaaattcga tgataagctg tcaaacatga 180
gaattggtcg acggcgcgcc aaagcttgca tgcctgcagc cgcgtaacct ggcaaaatcg 240
gttacggttg agtaataaat ggatgccctg cgtaagcggg gcacatttca ttacctcttt 300
ctccgcaccc gacatagata ataacttcgt atagtataca ttatacgaag ttatctagta 360
gacttaatta aggatcgatc cggcgcgcca atagtcatgc cccgcgccca ccggaaggag 420
ctgactgggt tgaaggctct caagggcatc ggtcgagctt ctatgcggca tcagagcaga 480
ttgtactgag agtgcaccat 500
<210> 66
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 66
tgcgtaagcg gggcacattt cattacctct ttctccgcac gctctgccag tgttacaacc 60
<210> 67
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 67
taatgtatac tatacgaagt tattatctat gtcgggtgct aacgcggtat gaaaatggat 60
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБ РЕДАКТИРОВАНИЯ ДНК В БЕСКЛЕТОЧНОЙ СИСТЕМЕ | 2019 |
|
RU2766717C1 |
| СПОСОБ АМПЛИФИКАЦИИ КОЛЬЦЕВОЙ ДНК | 2017 |
|
RU2748736C2 |
| СПОСОБ РЕПЛИКАЦИИ ИЛИ АМПЛИФИКАЦИИ КОЛЬЦЕВОЙ ДНК | 2018 |
|
RU2752611C2 |
| ОПОСРЕДОВАННАЯ НУКЛЕАЗОЙ СБОРКА ДНК | 2015 |
|
RU2707911C2 |
| КОЛЬЦЕВАЯ МОЛЕКУЛА ДНК, ХАРАКТЕРИЗУЮЩАЯСЯ ЗАВИСИМОЙ ОТ УСЛОВИЙ ТОЧКОЙ НАЧАЛА РЕПЛИКАЦИИ, СПОСОБ ИХ ПОЛУЧЕНИЯ И ИХ ПРИМЕНЕНИЕ В ГЕННОЙ ТЕРАПИИ | 2003 |
|
RU2307870C2 |
| СПОСОБ АМПЛИФИКАЦИИ ДНК НА ОСНОВЕ ВНЕДРЕНИЯ В ЦЕПЬ | 2015 |
|
RU2729983C2 |
| СИНТЕЗ ДНК | 2015 |
|
RU2714253C2 |
| СПОСОБ АМПЛИФИКАЦИИ ДНК И КОМПОЗИЦИЯ ДЛЯ ЕГО ОСУЩЕСТВЛЕНИЯ | 2000 |
|
RU2260055C2 |
| ТЕРМОСТАБИЛЬНАЯ ДНК-ПОЛИМЕРАЗА, СПОСОБ ЕЕ ПОЛУЧЕНИЯ, ФРАГМЕНТ ВЫДЕЛЕННОЙ ДНК, СПОСОБ АМПЛИФИКАЦИИ ДНК, СПОСОБ ВВЕДЕНИЯ МЕТКИ В ДНК И СПОСОБ ОБРАТНОЙ ТРАНСКРИПЦИИ | 1997 |
|
RU2197524C2 |
| СПОСОБЫ АМПЛИФИКАЦИИ ДНК ДЛЯ СОХРАНЕНИЯ СТАТУСА МЕТИЛИРОВАНИЯ | 2018 |
|
RU2754038C2 |
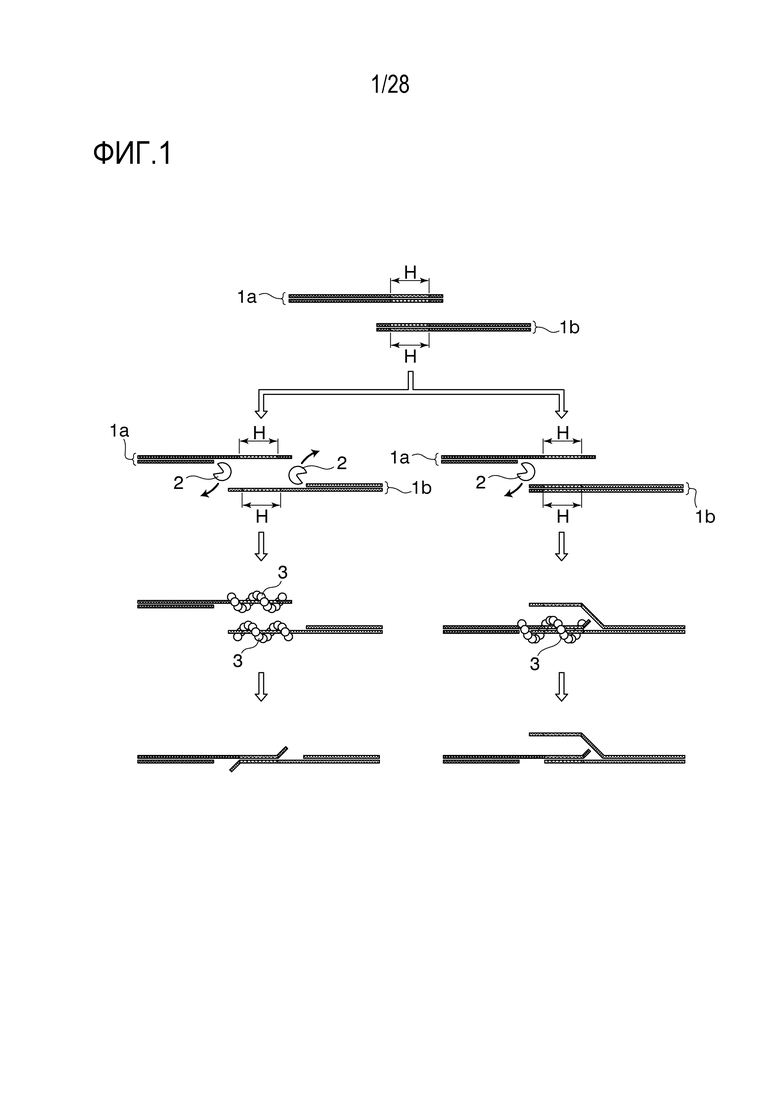
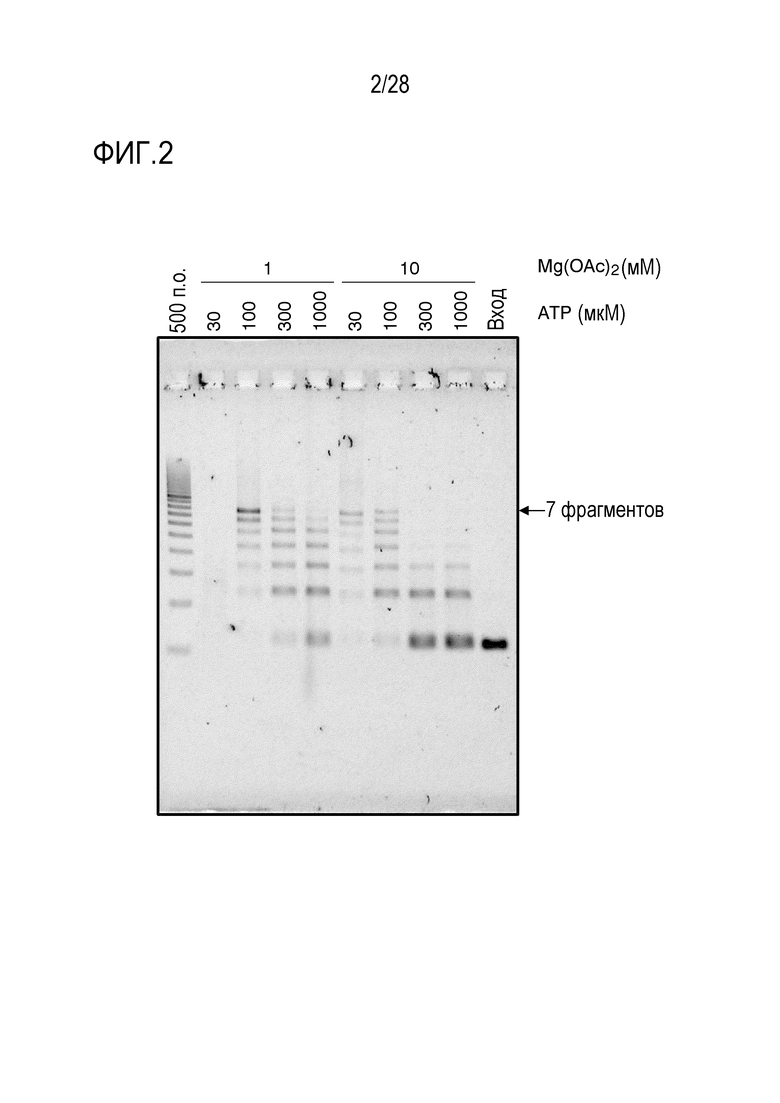
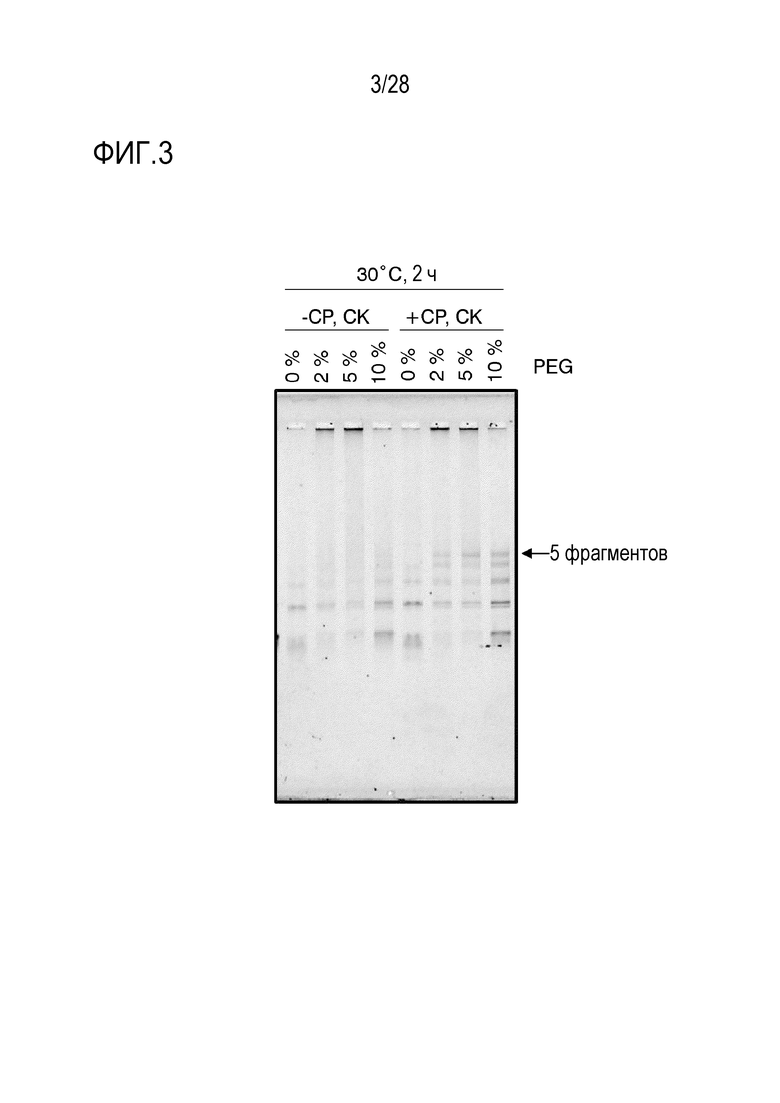
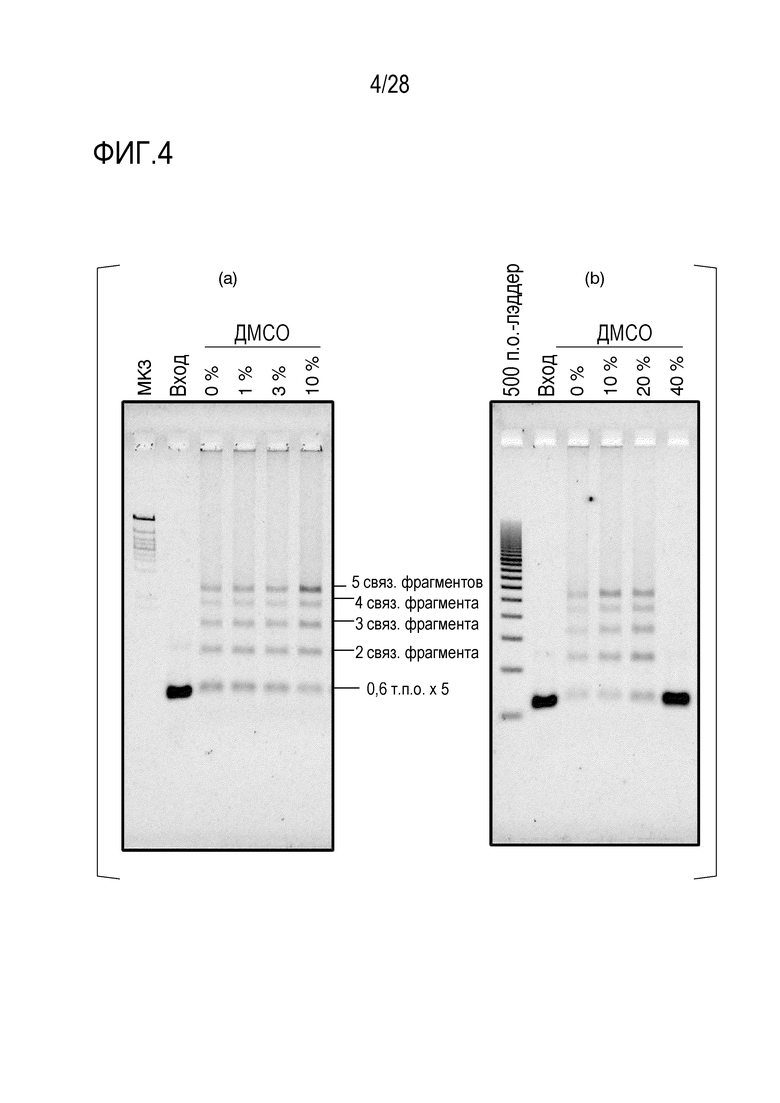
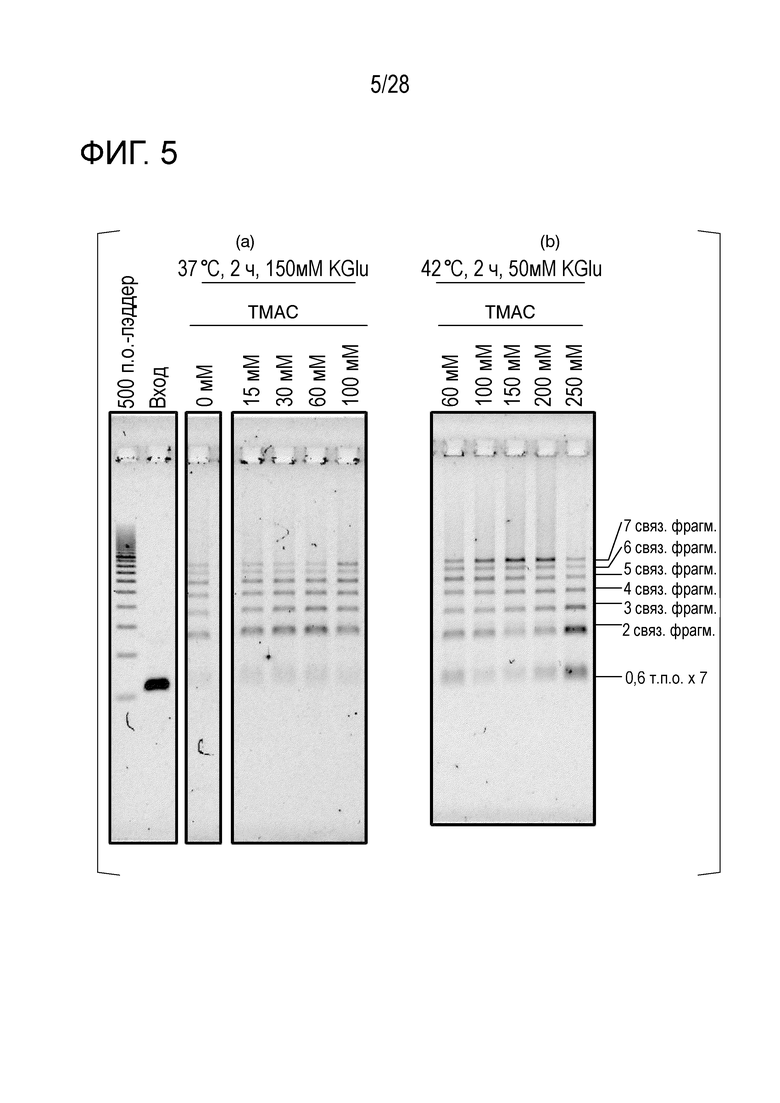
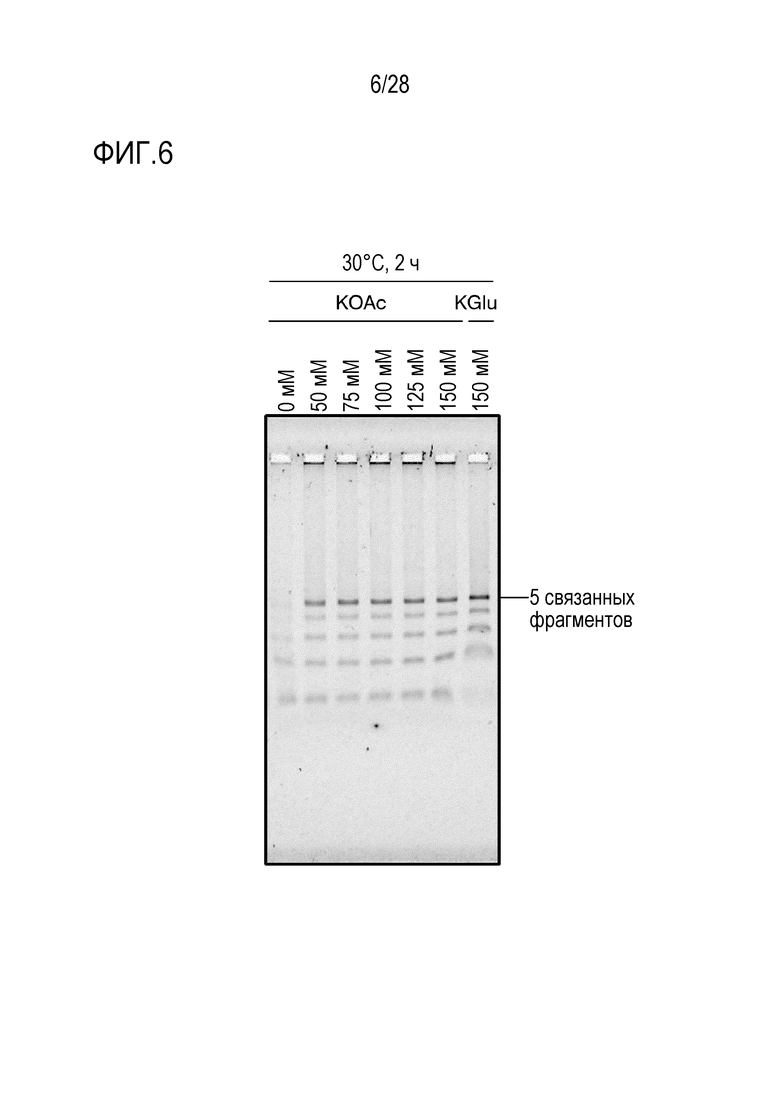
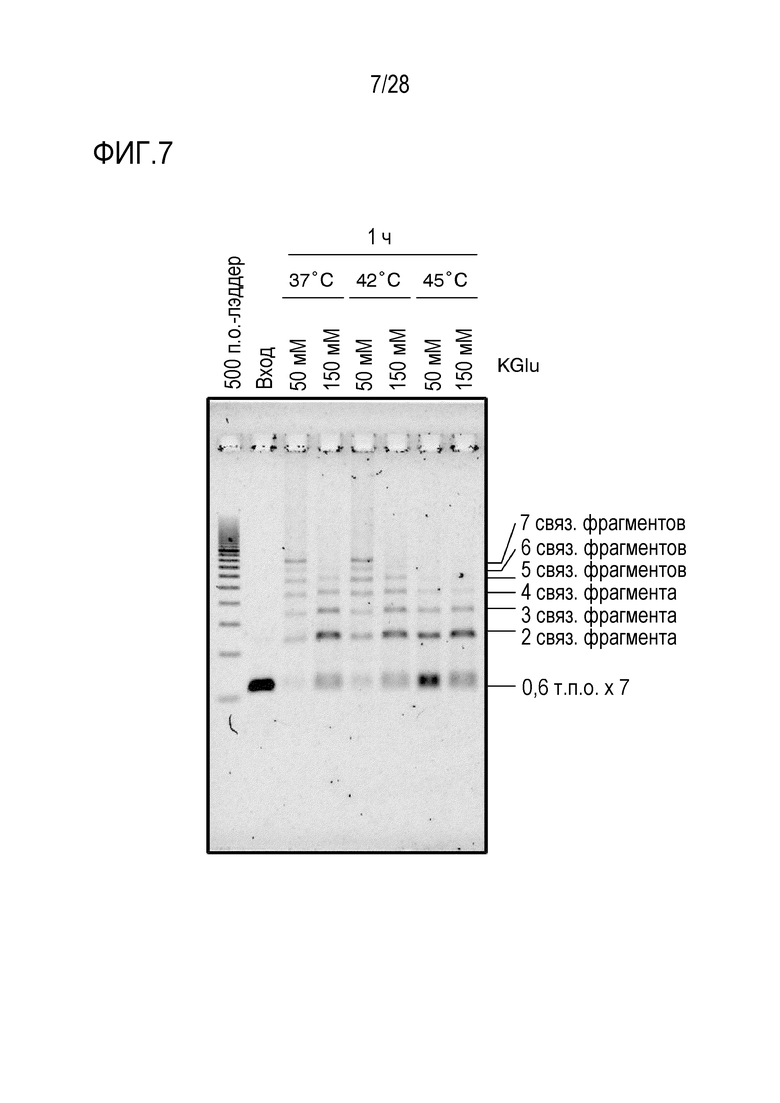
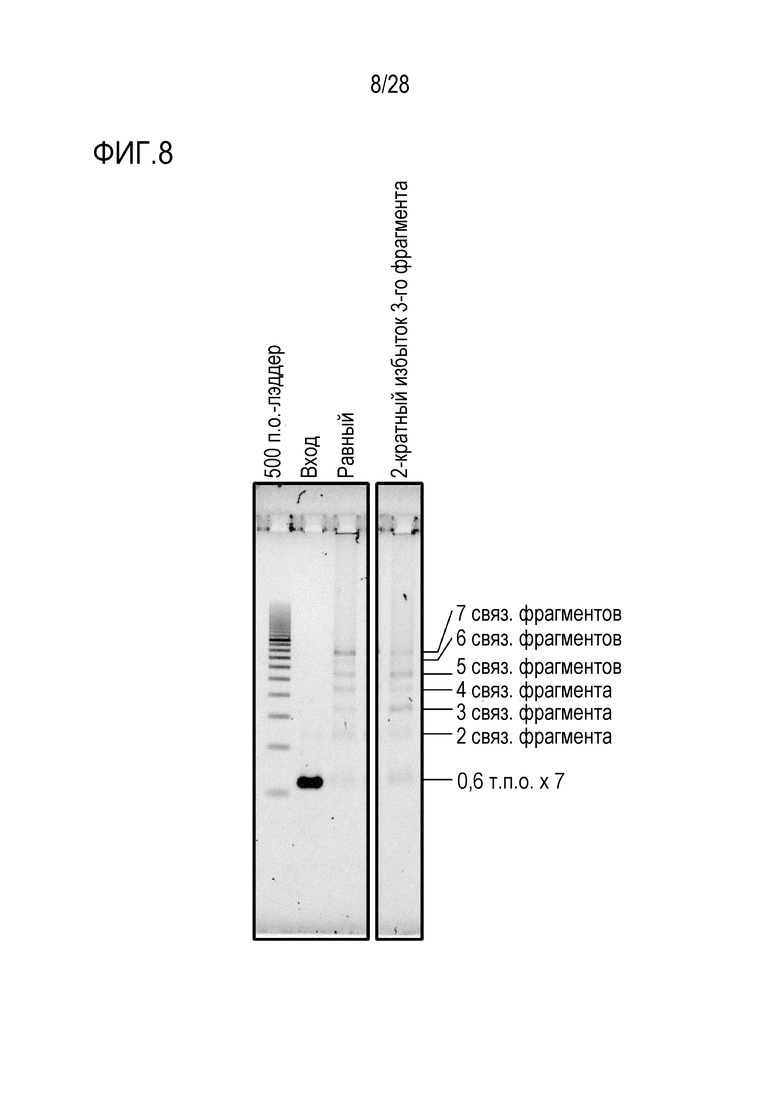
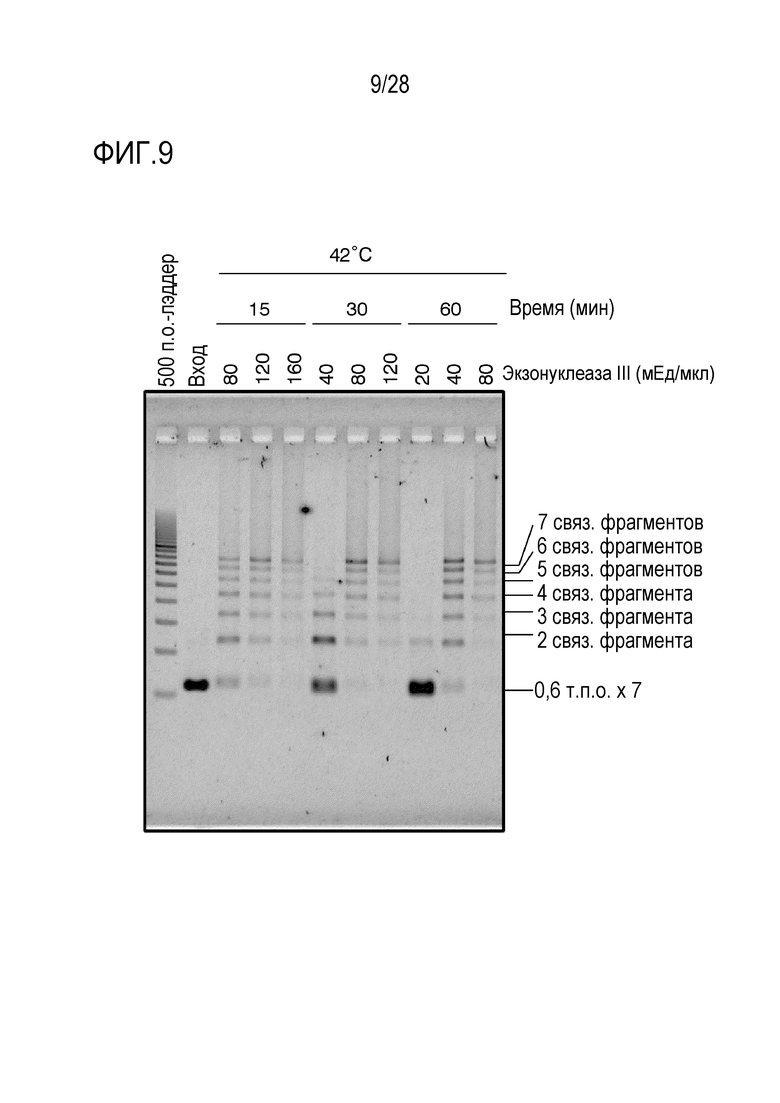
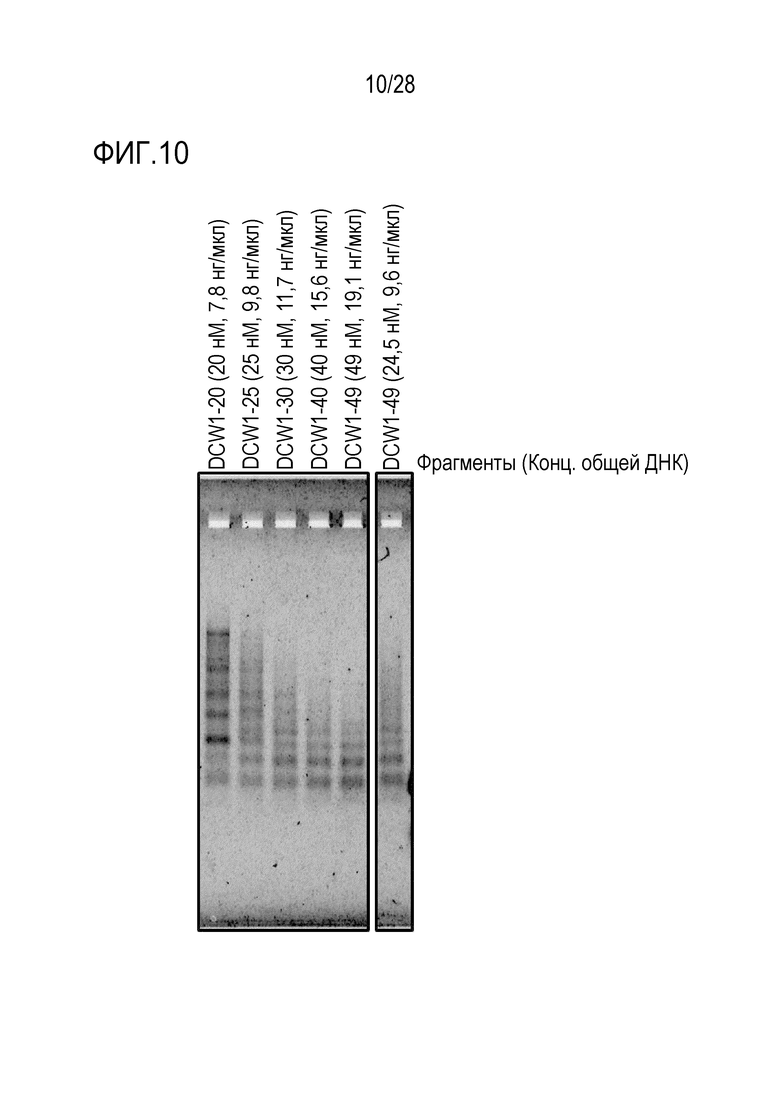
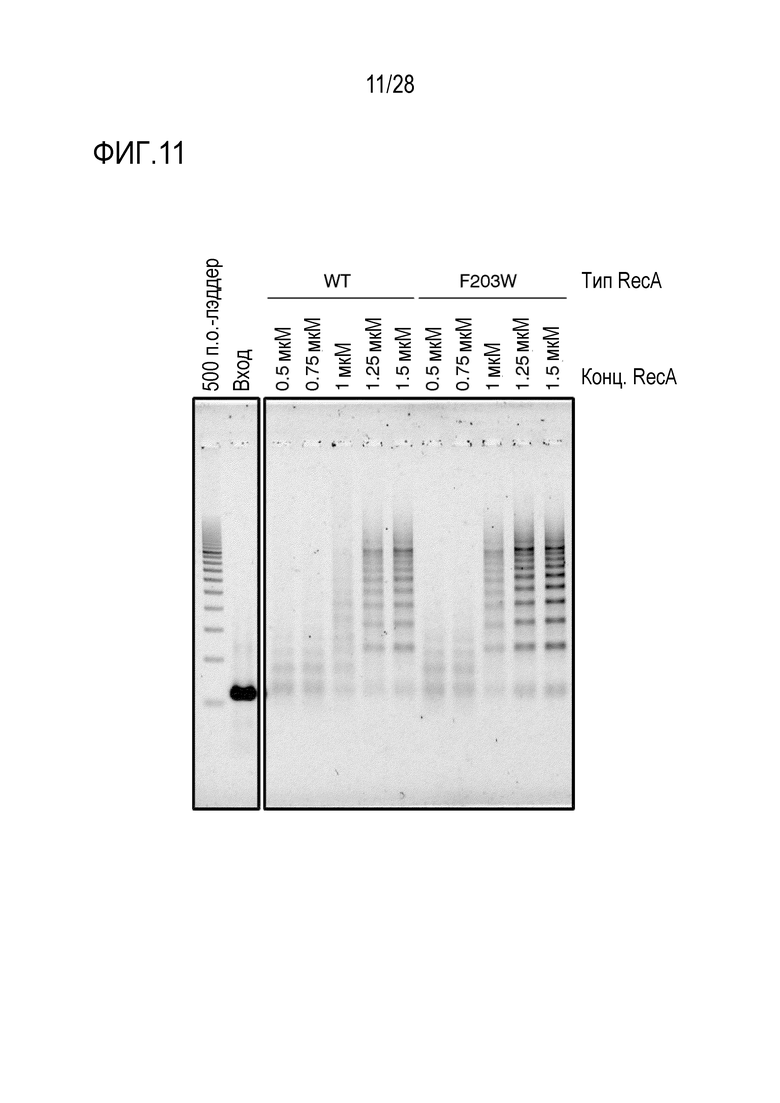
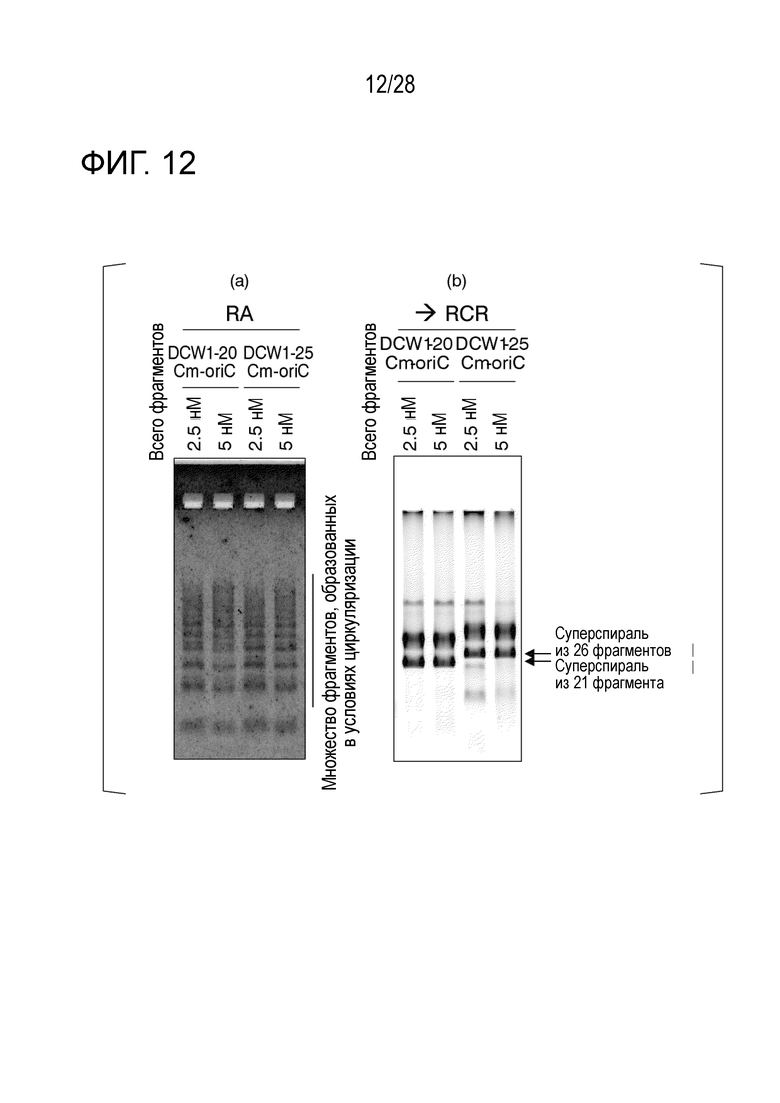
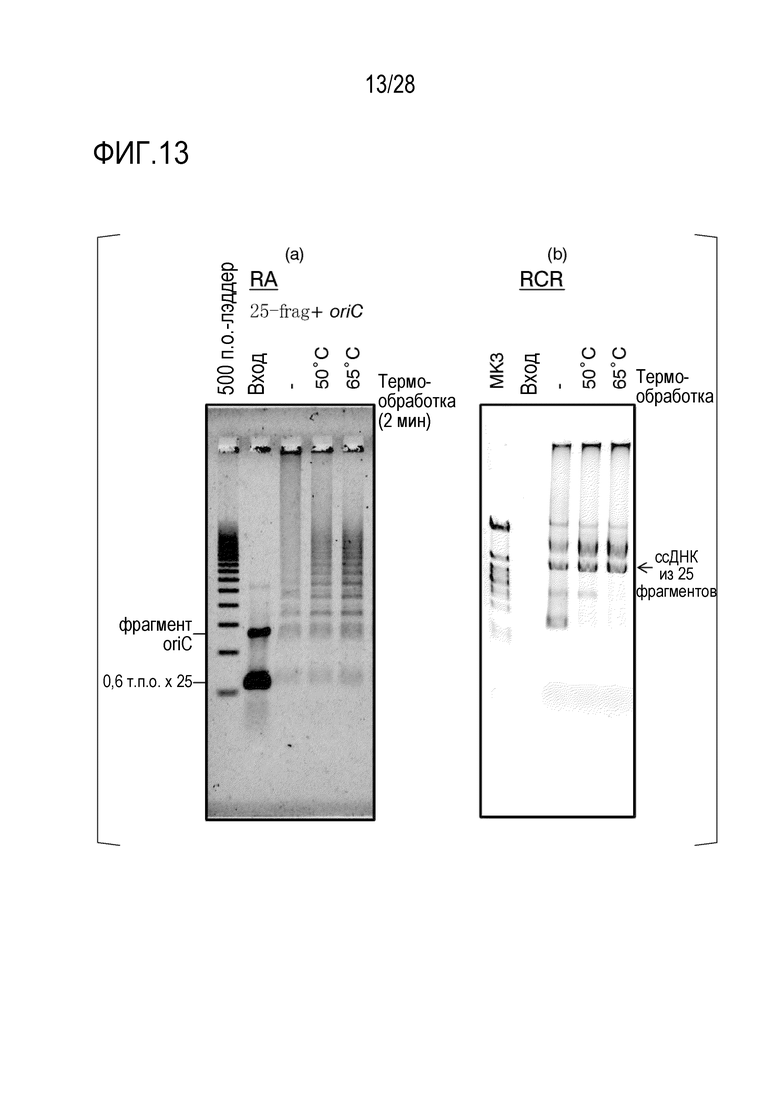
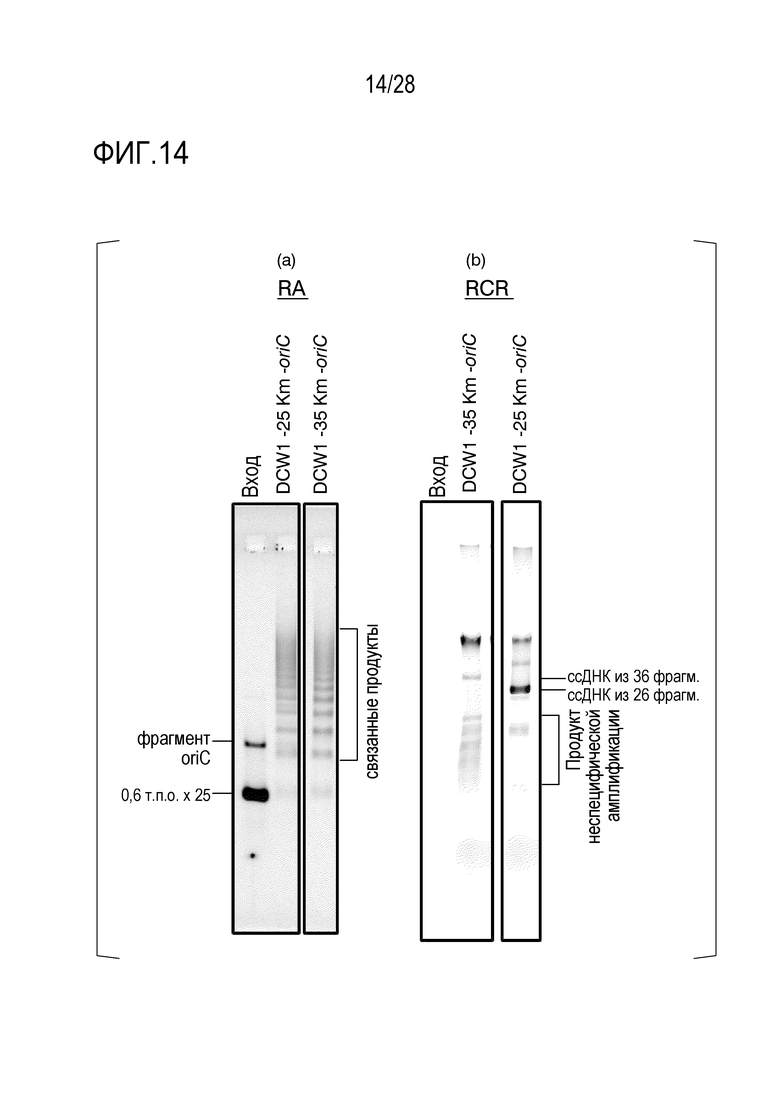
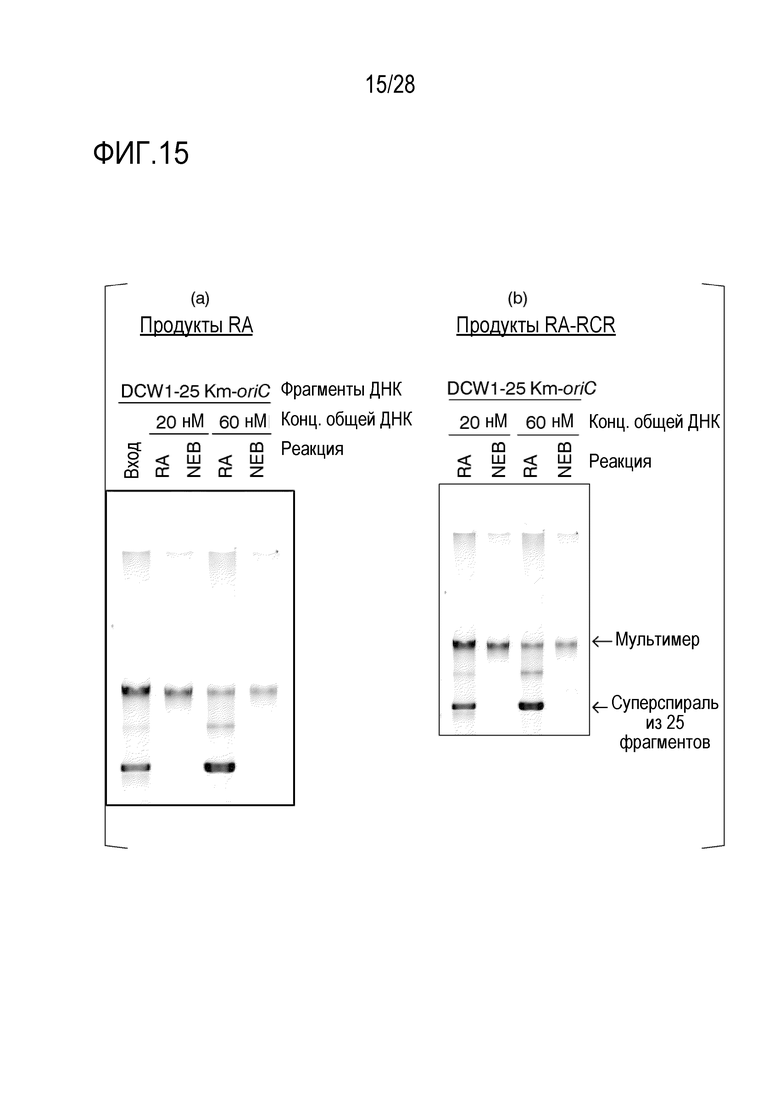
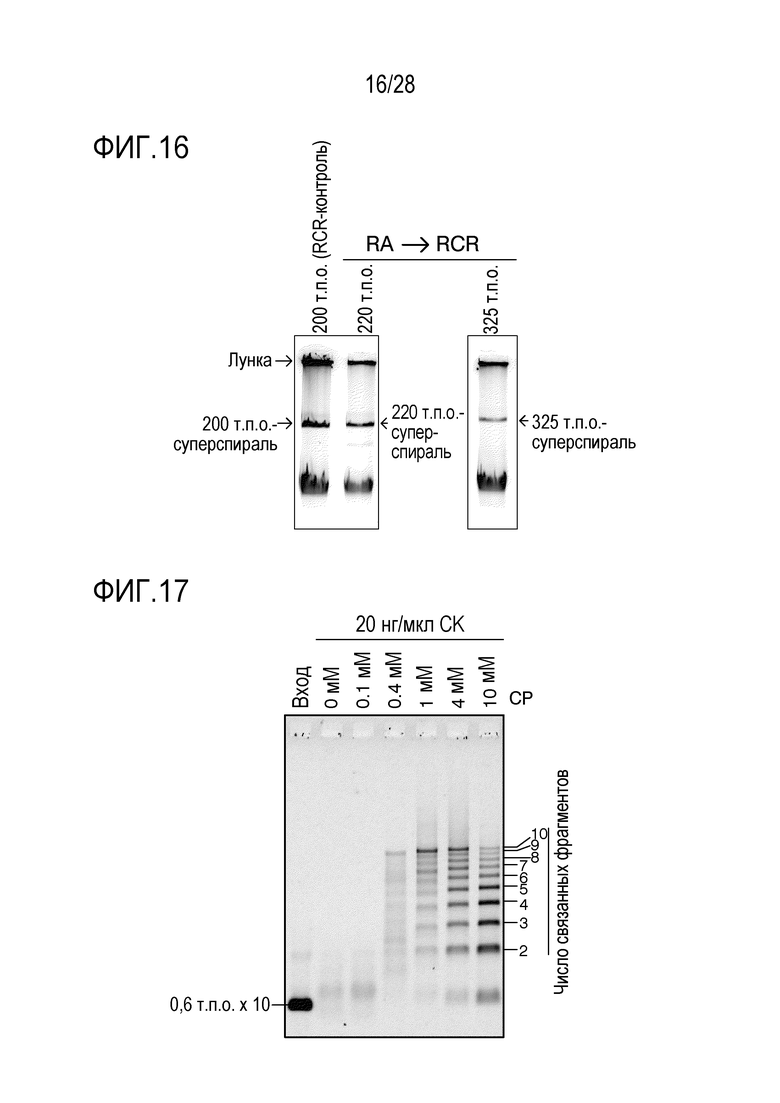
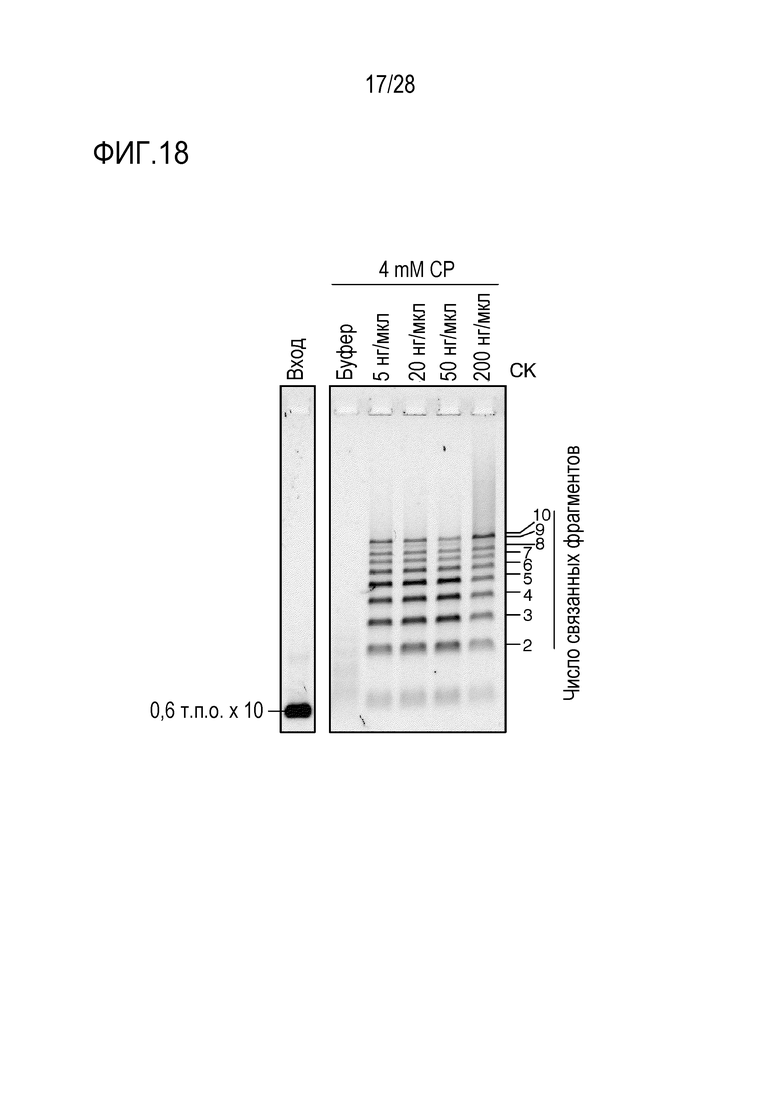
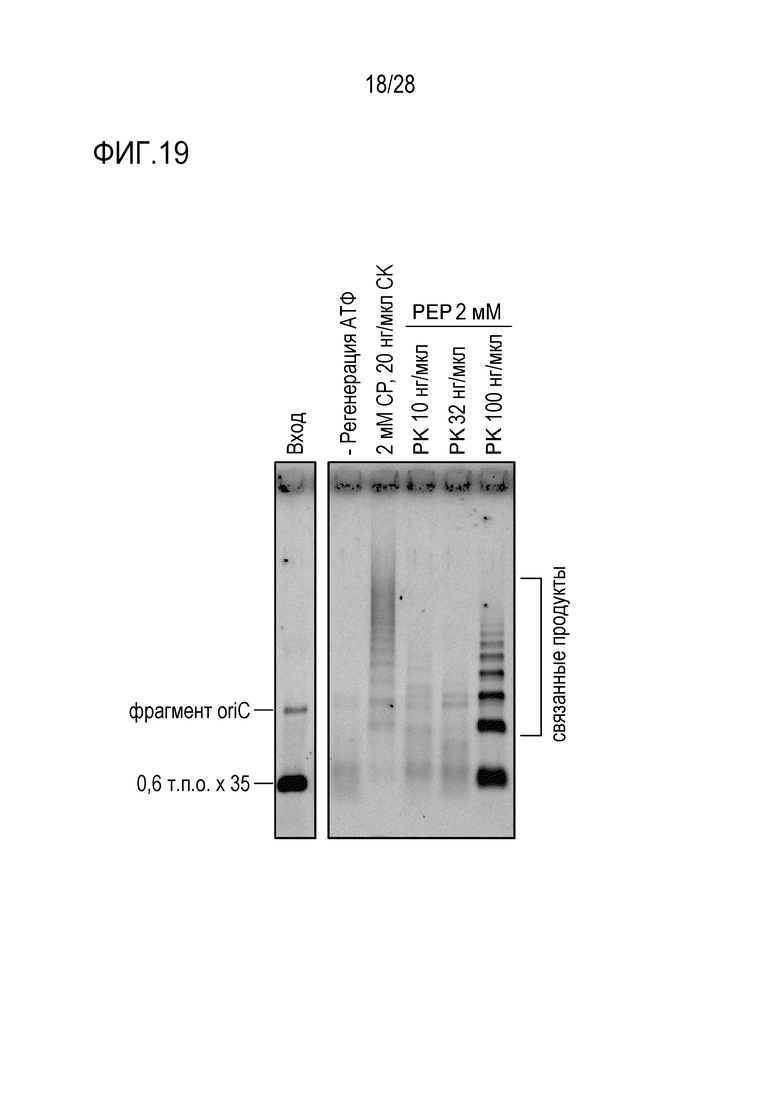
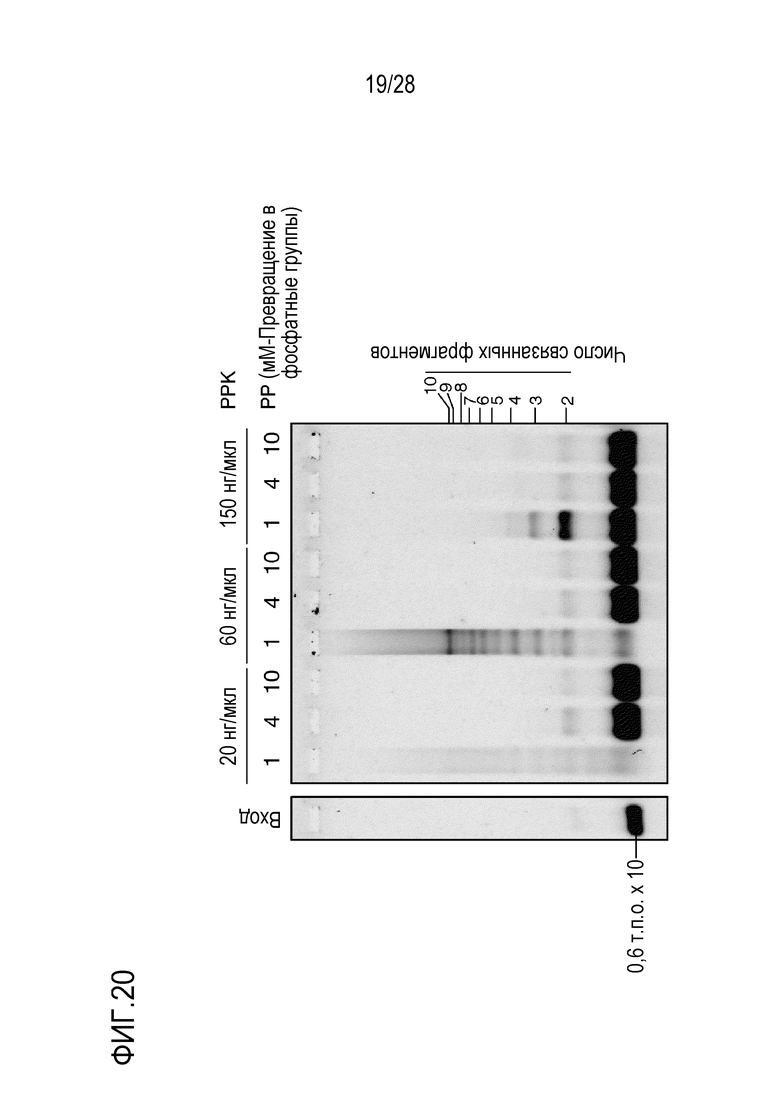
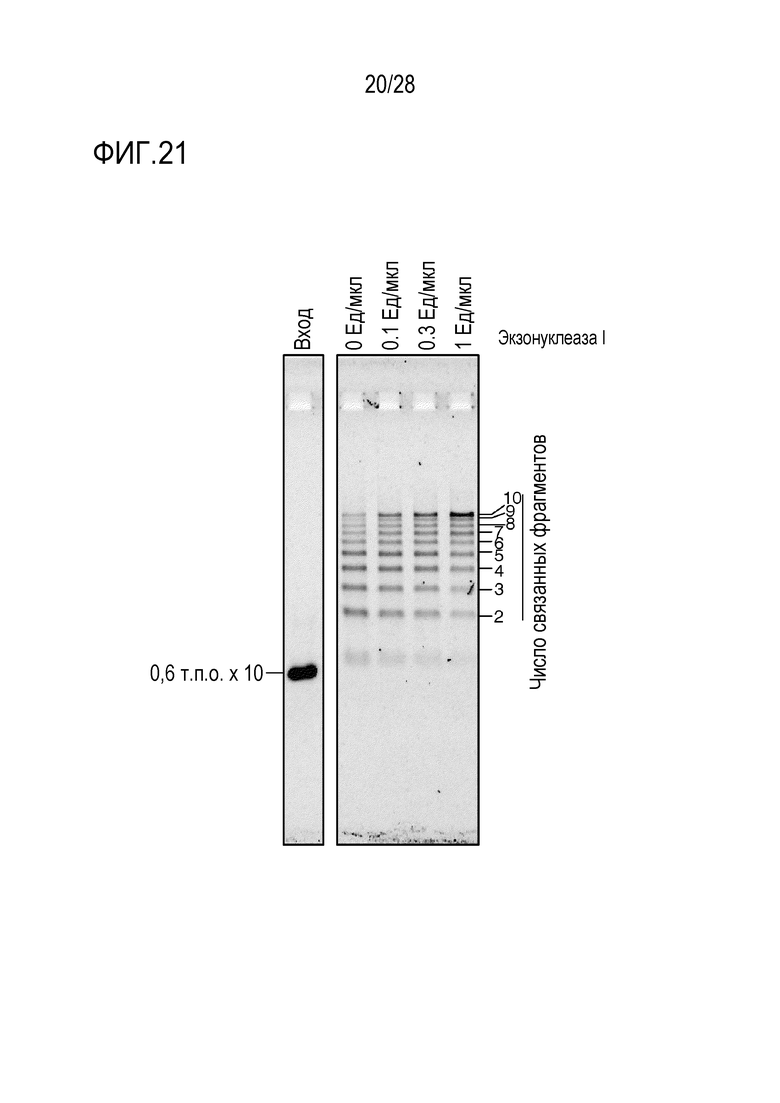
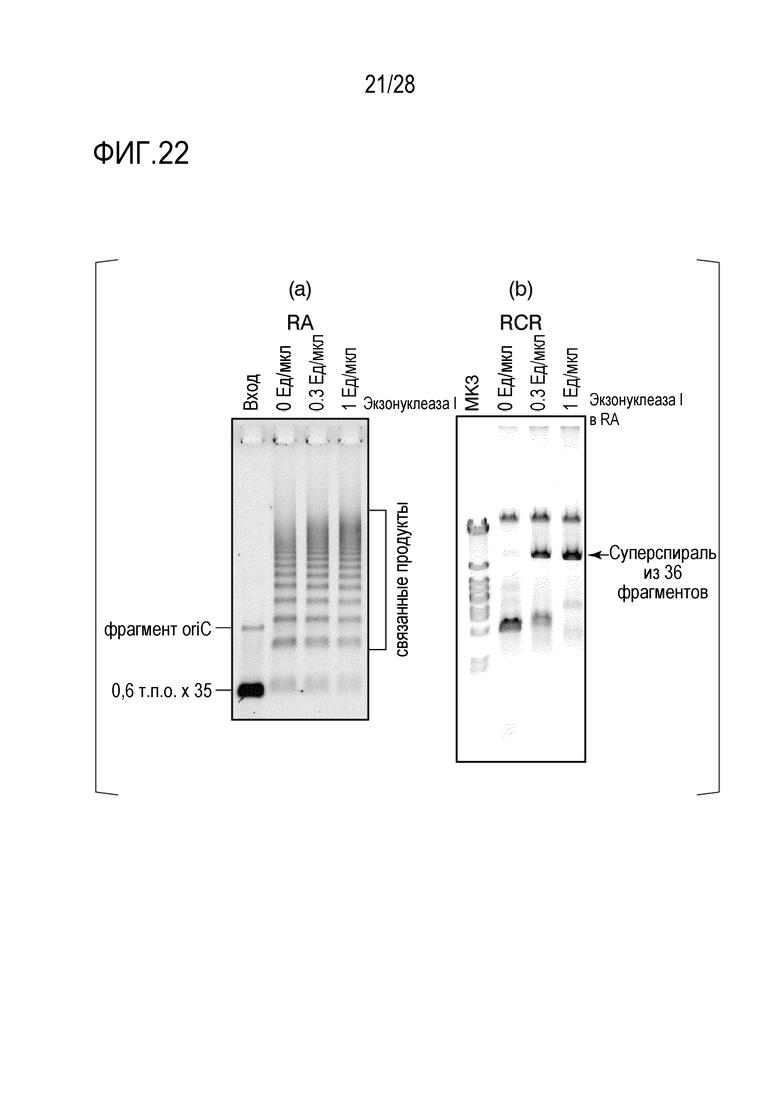
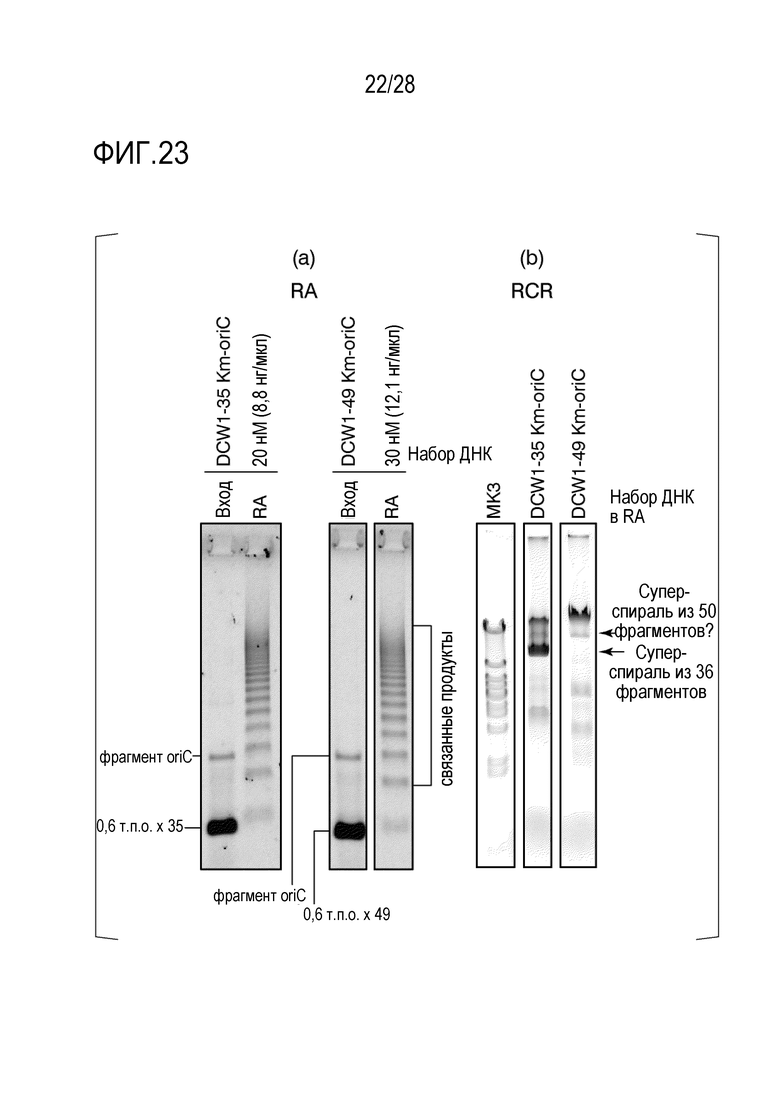
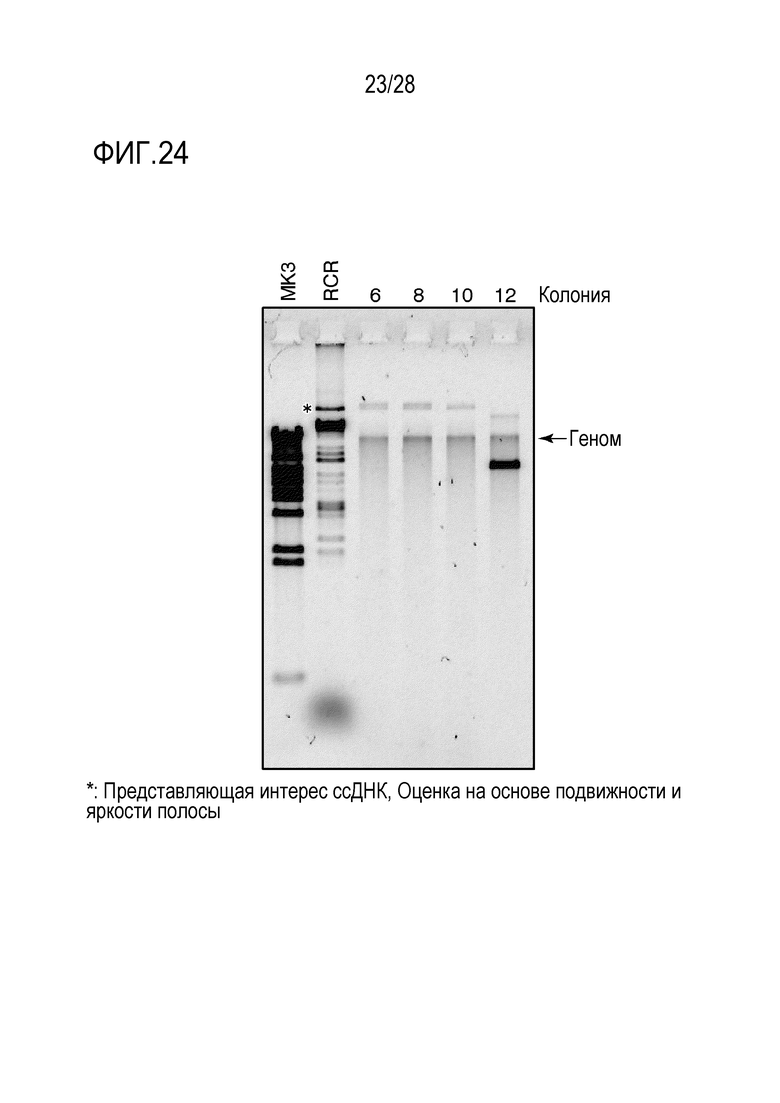
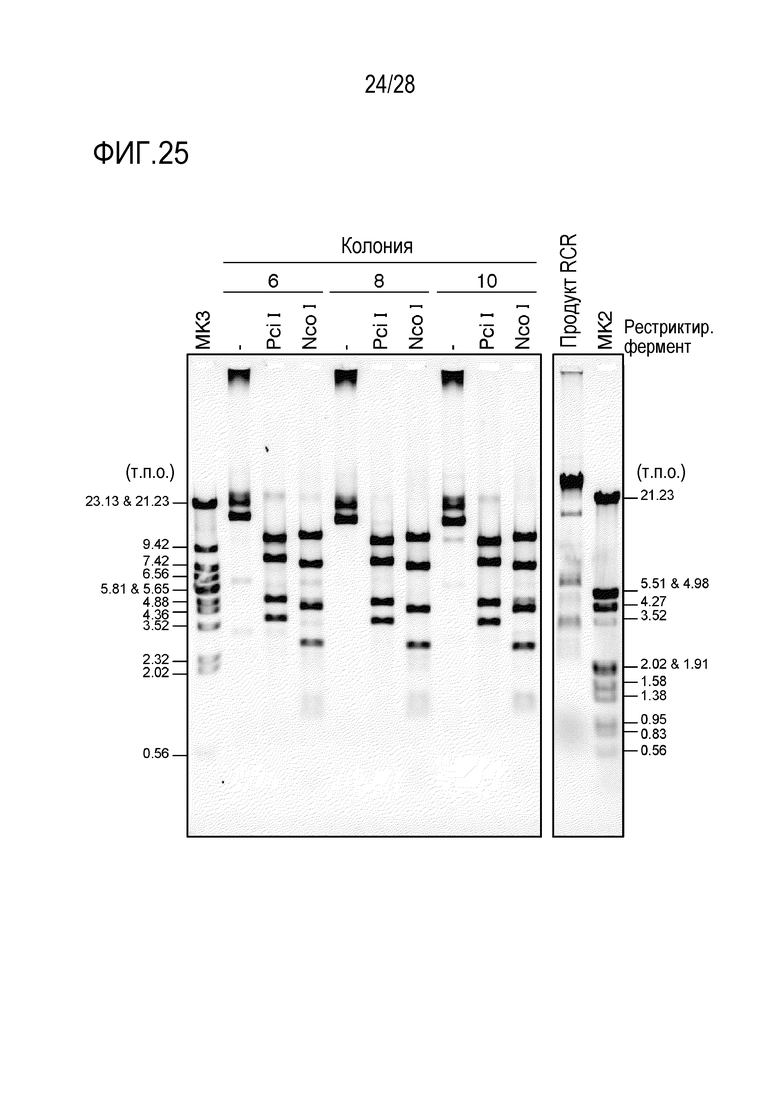
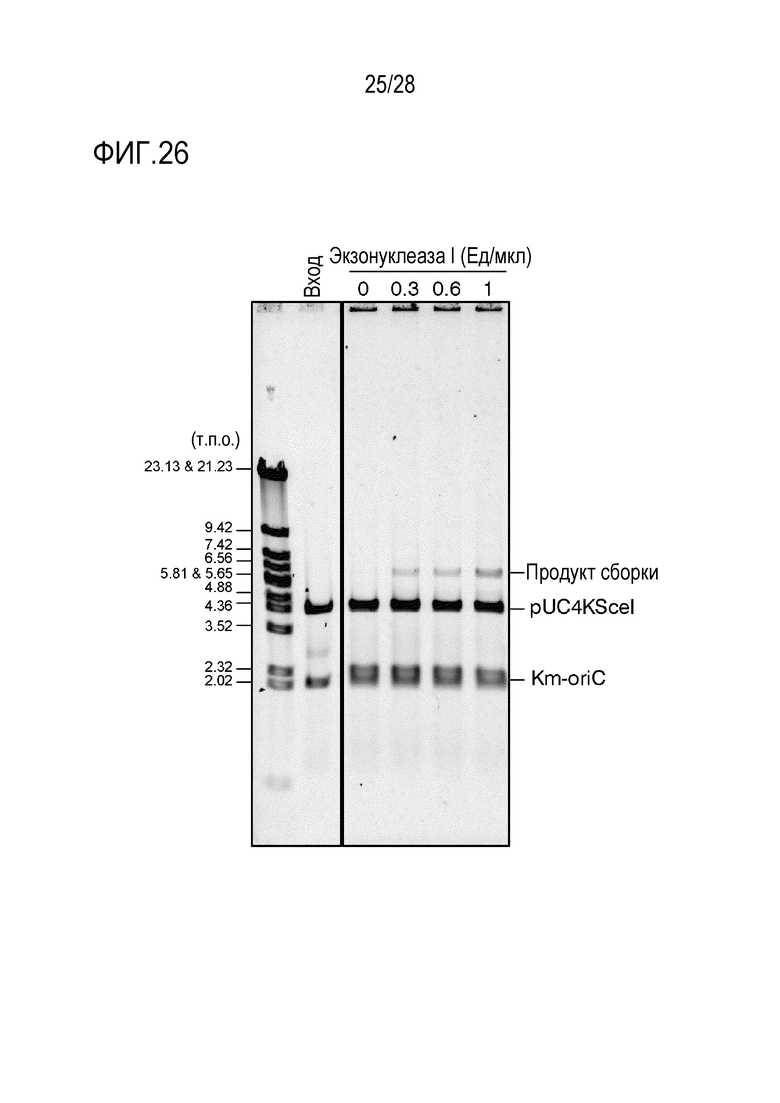
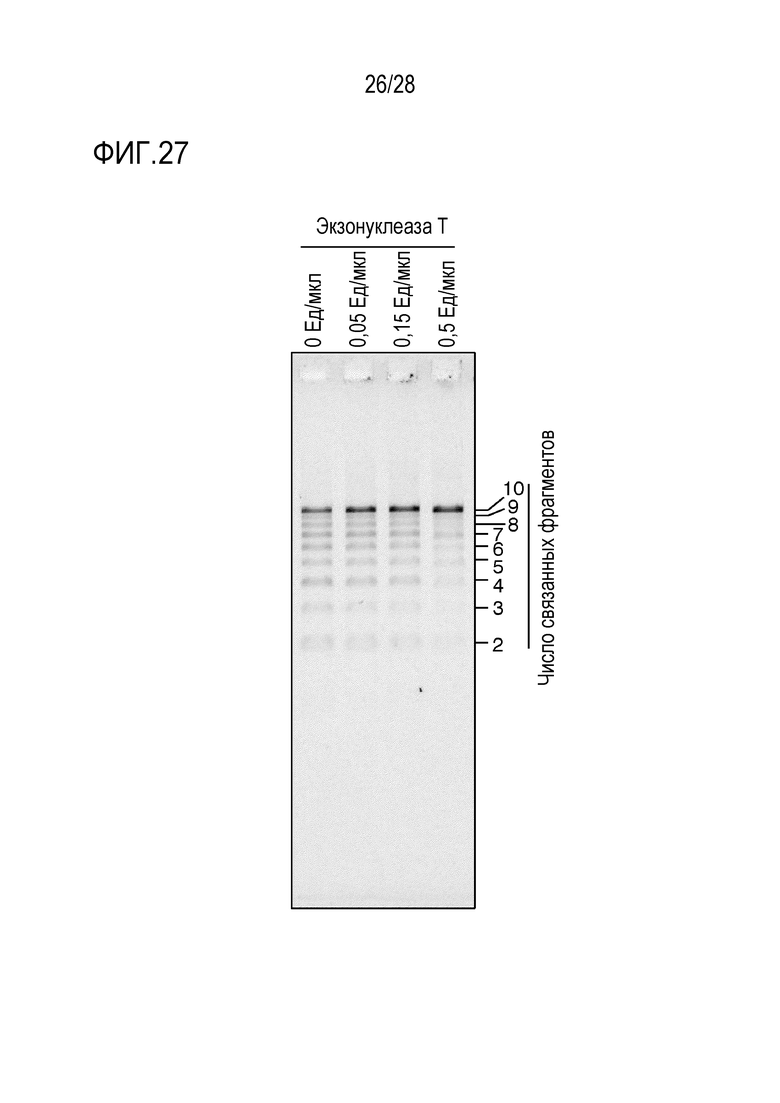
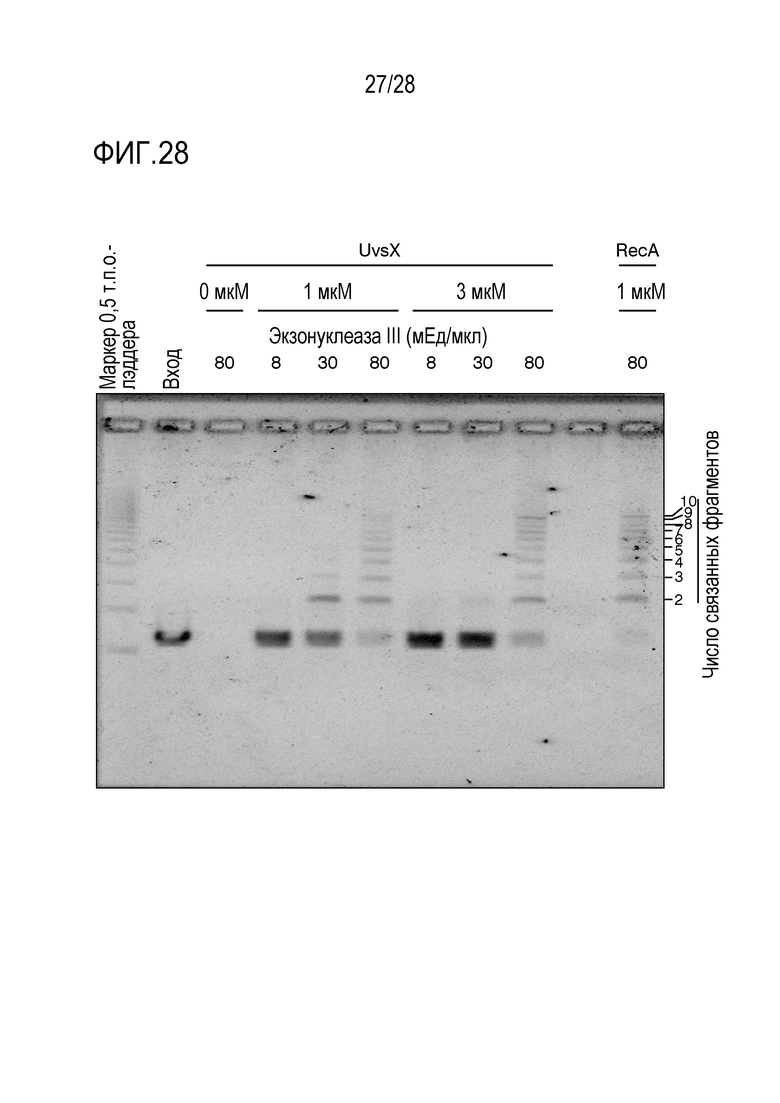
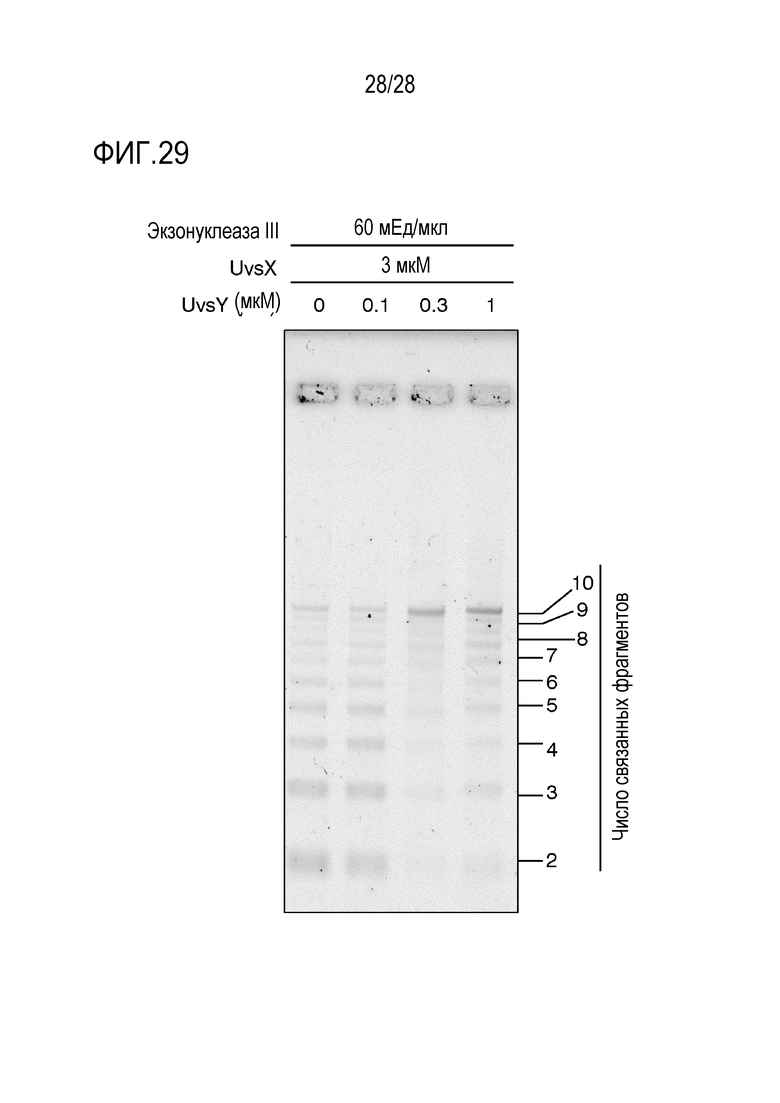
Настоящее изобретение относится к биотехнологии, а именно к способу получения линейной или кольцевой ДНК и к набору для связывания фрагментов ДНК, используемому в этом способе. Осуществляют получение линейной или кольцевой ДНК в реакционном растворе путем связывания фрагментов ДНК двух или более типов друг с другом в областях, имеющих гомологичные последовательности оснований, или в областях, имеющих комплементарные последовательности оснований, за одну реакцию. При этом области, имеющие гомологичные последовательности оснований, или области, имеющие комплементарные последовательности оснований, присутствуют в конце ДНК фрагмента или рядом с ним и имеют длину 20 п.о. или более. Реакцию связывания фрагментов ДНК двух или более типов осуществляют при температуре в пределах 25-48°C. Реакционный раствор содержит регенерирующий фермент для нуклеозид-трифосфатов или дезоксинуклеотид-трифосфатов и его субстрат. Способ позволяет эффективно связывать ДНК двух или более типов с использованием рекомбиназы семейства RecA и экзонуклеазы. 2 н. и 14 з.п. ф-лы, 29 ил., 1 табл., 25 пр.
1. Способ получения ДНК, где указанный способ включает:
получение реакционного раствора, содержащего фрагменты линейной двухцепочечной ДНК двух или более типов, белок, обладающий рекомбиназной активностью семейства RecA, и 3’→ 5’-экзонуклеазу, специфичную к линейной двухцепочечной ДНК, и
получение линейной или кольцевой ДНК в реакционном растворе путем связывания фрагментов ДНК двух или более типов друг с другом в областях, имеющих гомологичные последовательности оснований, или в областях, имеющих комплементарные последовательности оснований, за одну реакцию,
где области, имеющие гомологичные последовательности оснований, или области, имеющие комплементарные последовательности оснований, присутствуют в конце ДНК фрагмента или рядом с ним,
где области, имеющие гомологичные последовательности оснований, или области, имеющие комплементарные последовательности оснований, имеют длину 20 п.о. или более,
где реакцию связывания фрагментов ДНК двух или более типов осуществляют при температуре в пределах 25-48°C, и
где реакционный раствор содержит регенерирующий фермент для нуклеозид-трифосфатов или дезоксинуклеотид-трифосфатов и его субстрат.
2. Способ получения ДНК по п. 1, где реакционный раствор дополнительно содержит 3’→5’-экзонуклеазу, специфичную к одноцепочечной ДНК.
3. Способ получения ДНК по любому из пп. 1 и 2, где комбинация регенерирующего фермента и его субстрата представляет собой по меньшей мере одну комбинацию, выбранную из группы, состоящей из комбинации креатинкиназы и фосфата креатина, комбинации пируват-киназы и пирувата фосфоэнола, комбинации ацетат-киназы и ацетилфосфата, комбинации полифосфат-киназы и полифосфата и комбинации нуклеозид-дифосфаткиназы и нуклеозид-трифосфата.
4. Способ получения ДНК по любому из пп. 1-3, где линейную или кольцевую ДНК получают путем связывания 7 или более фрагментов ДНК.
5. Способ получения ДНК по любому из пп. 1-4, где реакционный раствор содержит одно или более веществ, выбранных из группы, состоящей из хлорида тетраметиламмония и диметилсульфоксида.
6. Способ получения ДНК по любому из пп. 1-5, где белком, обладающим рекомбиназной активностью семейства RecA, является uvsX, и реакционный раствор дополнительно содержит uvsY.
7. Способ получения ДНК по п. 6, где области, имеющие гомологичные последовательности оснований, имеют длину от 20 п.о. до 500 п.о.
8. Способ получения ДНК по любому из пп. 1-7, где реакционный раствор в начале реакции связывания фрагментов ДНК двух или более типов содержит фрагменты ДНК двух или более типов в одной и той же молярной концентрации.
9. Способ получения ДНК по любому из пп. 1-8, дополнительно включающий репарацию гэпов и ников в полученной линейной или кольцевой ДНК с использованием ферментов для репарации гэпов.
10. Способ получения ДНК по п. 9, дополнительно включающий термообработку полученной линейной или кольцевой ДНК при 50-70°С с последующим быстрым охлаждением до температуры 10°С или менее, а затем репарацию гэпов и ников с использованием ферментов для репарации гэпов.
11. Способ получения ДНК по любому из пп. 1-8, где ДНК, полученная путем связывания, является линейной, а ПЦР проводят непосредственно с использованием линейной ДНК в качестве матрицы.
12. Способ получения ДНК по любому из пп. 1-8, где ДНК, полученная путем связывания, является кольцевой ДНК, содержащей последовательность ориджина репликации, способную связываться с ферментом, обладающим активностью DnaA, и
образовывать реакционную смесь, содержащую кольцевую ДНК, первую группу ферментов, которые катализируют репликацию кольцевой ДНК; вторую группу ферментов, которые катализируют реакцию связывания фрагментов Оказаки и синтезируют две сестринских кольцевых ДНК, состоящих из катенана; третью группу ферментов, которые катализируют разделение двух сестринских кольцевых ДНК, и dNTP.
13. Способ получения ДНК по любому из пп. 1-8, дополнительно включающий введение полученной линейной или кольцевой ДНК в микроорганизм и амплификацию двухцепочечной ДНК с репарацией гэпов и ников.
14. Набор для связывания фрагментов ДНК, содержащий:
белок, обладающий рекомбиназной активностью семейства RecA, 3’→ 5’-экзонуклеазу, специфичную к линейной двухцепочечной ДНК, и регенерирующий фермент для нуклеозид-трифосфатов или дезоксинуклеотид-трифосфатов и его субстрат,
где указанный набор используют для получения линейной или кольцевой ДНК посредством связывания фрагментов линейной двухцепочечной ДНК двух или более типов друг с другом в областях, имеющих гомологичные последовательности оснований, или областях, имеющих комплементарные последовательности оснований,
где области, имеющие гомологичные последовательности оснований, или области, имеющие комплементарные последовательности оснований, присутствуют в конце ДНК фрагмента или рядом с ним, и
где области, имеющие гомологичные последовательности оснований, или области, имеющие комплементарные последовательности оснований, имеют длину 20 п.о. или более.
15. Набор для связывания фрагментов ДНК по п. 14, дополнительно содержащий 3’→ 5’-экзонуклеазу, специфичную к одноцепочечной ДНК.
16. Набор для связывания фрагментов ДНК по п. 14 или 15, где комбинация регенерирующего фермента и его субстрата представляет собой по меньшей мере одну комбинацию, выбранную из группы, состоящей из комбинации креатинкиназы и фосфата креатина, комбинации пируват-киназы и пирувата фосфоэнола, комбинации ацетат-киназы и ацетилфосфата, комбинации полифосфат-киназы и полифосфата и комбинации нуклеозид-дифосфаткиназы и нуклеозид-трифосфата.
| Пресс для выдавливания из деревянных дисков заготовок для ниточных катушек | 1923 |
|
SU2007A1 |
| Токарный резец | 1924 |
|
SU2016A1 |
| WO 2016080424 A1, 26.05.2016 | |||
| ГОНЧАРЕНКО Г.Г | |||
| Основы генетической инженерии | |||
| Методическое пособие, Гомель, 2003, с | |||
| Приспособление для плетения проволочного каркаса для железобетонных пустотелых камней | 1920 |
|
SU44A1 |
Авторы
Даты
2022-04-01—Публикация
2018-07-05—Подача