ПЕРЕКРЕСТНЫЕ ССЫЛКИ НА РОДСТВЕННЫЕ ЗАЯВКИ
Настоящая заявка испрашивает приоритет согласно §119 (e) Раздела 35 Свода законов США на основании предварительной заявки на патент США №62/414 279, поданной 28 октября 2016 г., и предварительной заявки на патент США №62/385 079, поданной 8 сентября 2016 г., каждая из которых в полном объеме включена в настоящую заявку посредством ссылки.
ЗАЯВЛЕНИЕ ОТНОСИТЕЛЬНО ПЕРЕЧНЯ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
Перечень последовательностей, связанный с данной заявкой, предоставлен в текстовом формате вместо бумажной копии и тем самым включен посредством ссылки в данное описание. Название текстового файла, содержащего перечень последовательностей, - BLBD_076_02WO_ST25.txt. Размер текстового файла составляет 266 кБ, он был создан 8 сентября 2017 года и подан в электронном виде через EFS-Web одновременно с подачей данного описания.
Уровень техники
Область техники
Настоящее изобретение относится к улучшенным композициям для редактирования генома. В частности, изобретение относится к вариантам нуклеаз, композициям и способам их применения для редактирования гена запрограммированной клеточной гибели человека 1 (PD-1).
Описание предшествующего уровня техники
Глобальное бремя рака удвоилось в период между 1975 и 2000 годами. Рак является второй по значимости причиной заболеваемости и смертности во всем мире, с приблизительно 14,1 миллиона новых случаев заболевания и 8,2 миллиона смертей от рака с 2012 году. Наиболее распространенными видами рака являются рак молочной железы, рак легких и бронхов, рак простаты, рак толстой и прямой кишки, рак мочевого пузыря, меланома кожи, неходжкинская лимфома, рак щитовидной железы, рак почки и почечной лоханки, рак эндометрия, лейкоз и рак поджелудочной железы. Увеличение числа новых случаев заболевания раком прогнозируется до 22 миллионов в течение следующих двух десятилетий.
Иммунная система играет ключевую роль в выявлении и борьбе с раком человека. Большинство трансформированных клеток быстро обнаруживаются иммунными стражами и разрушаются посредством активации антиген-специфических Т-клеток через клонально экспрессируемые Т-клеточные рецепторы (TCR). Соответственно, рак можно считать иммунологическим расстройством, неспособностью иммунной системы выработать необходимый противоопухолевый ответ для устойчивого подавления и устранения заболевания. В целях более эффективной борьбы с раком определенные иммунотерапевтические вмешательства, разработанные в последние несколько десятилетий, были специально направлены на повышение Т- клеточного иммунитета. Эти методы лечения приводили только к спорадическим случаям ремиссии заболевания и не имели существенного общего успеха. Более поздние методы лечения, с применением молекул, нацеленных на моноклональные антитела, которые ингибируют активацию Т-клеток, такие как CTLA-4 или PD-1, показали более существенный противоопухолевый эффект; однако эти методы лечения также ассоциированы с существенной токсичностью из-за системной иммунной активации.
Совсем недавно были изучены и испытаны на ранних стадиях клинических испытаний стратегии адоптивной клеточной иммунотерапии, основанные на выделении, модификации, экспансии и реинфузии Т-клеток. Т-клетки часто являются эффекторными клетками выбора для иммунотерапии рака благодаря их избирательному распознаванию и мощным эффекторным механизмам. Эти методы лечения продемонстрировали смешанные показатели успеха, но у небольшого числа пациентов наблюдалась длительная ремиссия, что указывает на пока еще не реализованный потенциал для иммунотерапии на основе Т-клеток.
Успешное распознавание антигенов, ассоциированных с опухолевыми клетками, цитолитическими Т-клетками инициирует направленный лизис опухоли и лежит в основе любого эффективного подхода иммунотерапии рака. Инфильтрирующие опухоль Т-клетки (TIL) экспрессируют TCR, специфически направленные на опухоль-ассоциированные антигены; тем не менее, наличие значительного количества TIL ограничено только несколькими видами рака человека. Сконструированные Т-клеточные рецепторы (TCR) и химерные антигенные рецепторы (CAR) потенциально увеличивают применимость иммунотерапии на основе Т-клеток для многих видов рака и других иммунных нарушений.
Кроме того, современные сконструированные Т-клетки все еще регулируются сложным иммунодепрессивным микроокружением, которое состоит из раковых клеток, воспалительных клеток, стромальных клеток и цитокинов. Среди этих компонентов раковые клетки, воспалительные клетки и супрессивные цитокины регулируют фенотип и функцию Т-клеток. В совокупности микроокружение опухоли заставляет Т-клетки терминально дифференцироваться в истощенные Т-клетки.
Истощение Т-клеток - это состояние дисфункции Т-клеток в хронической среде, которое характеризуется повышенной экспрессией ингибирующих рецепторов или усилением их сигналов; снижением выработки эффекторных цитокинов; и уменьшением способности выживать и устранять рак. Истощенные Т-клетки также демонстрируют потерю функции иерархическим образом: снижение выработки IL-2 и способность к уничтожению ex vivo теряются на ранней стадии истощения, выработка TNF-α теряется на промежуточной стадии, а выработка IFN-γ и GzmB теряются на поздней стадии истощения. Большинство Т-клеток в микроокружении опухоли дифференцируются в истощенные Т-клетки, теряют способность устранять рак и в конечном итоге выводятся из игры.
Белок запрограммированной гибели клеток 1 (PD-1) экспрессируется на Т-клетках и опосредует иммуносупрессию путем связывания с иммуносупрессивными факторами, например, PD-L1 и PD-L2, присутствующими в микроокружении опухоли. Экспрессия PD-L1 и PD-L2 коррелирует с прогнозом при некоторых злокачественных опухолях человека. Сигнальный путь PD-L1/PD-1 является одним из важных регуляторных путей истощения Т-клеток. PD-L1 широко экспрессируется в раковых клетках и стромальных клетках, а блокада PD-L1/PD-1 с помощью моноклональных антител усиливает противоопухолевую функцию Т-клеток. PD-L2 также связывается с PD-1 и отрицательно регулирует функцию Т-клеток.
Краткое описание
В целом настоящее изобретение относится, в частности, к композициям, содержащим варианты хоминг-эндонуклеазы и megaTAL, которые расщепляют сайт-мишень в гене PD-1 человека, и способам их применения.
В различных вариантах реализации настоящее изобретение, в частности, предусматривает полипептид, содержащий вариант хоминг-эндонуклеазы (HE), который расщепляет сайт-мишень в гене запрограммированной гибели клеток человека 1 (PD-1).
В частных вариантах реализации вариант HE представляет собой вариант LAGLIDADG хоминг-эндонуклеазы (LHE).
В некоторых вариантах реализации указанный полипептид содержит биологически активный фрагмент указанного варианта HE.
В некоторых вариантах реализации в указанном биологически активном фрагменте отсутствуют 1, 2, 3, 4, 5, 6, 7 или 8 N-концевых аминокислот по сравнению с соответствующей HE дикого типа.
В дополнительных вариантах реализации в биологически активном фрагменте отсутствуют 4 N-концевые аминокислоты по сравнению с соответствующей HE дикого типа.
В некоторых вариантах реализации в биологически активном фрагменте отсутствуют 8 N-концевых аминокислот по сравнению с соответствующей HE дикого типа.
В частных вариантах реализации в биологически активном фрагменте отсутствуют 1, 2, 3, 4 или 5 С-концевых аминокислот по сравнению с соответствующей HE дикого типа.
В частных вариантах реализации в биологически активном фрагменте отсутствует С-концевая аминокислота по сравнению с соответствующей HE дикого типа.
В некоторых вариантах реализации в биологически активном фрагменте отсутствуют 2 С-концевые аминокислоты по сравнению с соответствующей HE дикого типа.
В дополнительных вариантах реализации вариант HE представляет собой вариант LHE, выбранный из группы, состоящей из: I-AabMI, I-AaeMI, I-AniI, I-ApaMI, I-CapIII, I-CapIV, I-CkaMI, I-CpaMI, I-CpaMII, I-CpaMIII, I-CpaMIV, I-CpaMV, I-CpaV, I-CraMI, I-EjeMI, I-GpeMI, I-GpiI, I-GzeMI, I-GzeMII, I-GzeMIII, I-HjeMI, I-LtrII, I-LtrI, I-LtrWI, I-MpeMI, I-MveMI, I-NcrII, I-Ncrl, I-NcrMI, I-OheMI, I-OnuI, I-OsoMI, I-OsoMII, I-OsoMIII, I-OsoMIV, I-PanMI, I-PanMII, I-PanMIII, I-PnoMI, I-ScuMI, I-SmaMI, I-SscMI и I-Vdi141I.
В частных вариантах реализации вариант HE представляет собой вариант LHE, выбранный из группы, состоящей из: I-CpaMI, I-HjeMI, I-OnuI, I-PanMI и SmaMI.
В дополнительных вариантах реализации вариант HE представляет собой вариант I-OnuI LHE.
В дополнительных вариантах реализации вариант HE содержит одну или более аминокислотных замен на поверхностном участке распознавания ДНК в положениях аминокислот, выбранных из группы, состоящей из: 19, 24, 26, 28, 30, 32, 34, 35, 36, 37, 38, 40, 42, 44, 46, 48, 59, 68, 70, 72, 75, 76 77, 78, 80, 82, 168, 180, 182, 184, 186, 188, 189, 190, 191, 192, 193, 195, 197, 199, 201, 203, 223, 225, 227, 229, 231, 232, 234, 236, 238 и 240 аминокислотной последовательности I-OnuI LHE, представленной в SEQ ID NO: 1-5, или соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант HE содержит по меньшей мере 5, по меньшей мере 15, предпочтительно по меньшей мере 25, более предпочтительно по меньшей мере 35 или даже более предпочтительно по меньшей мере 40 или более аминокислотных замен в интерфейсе распознавания ДНК в положениях аминокислот, выбранных из группы, состоящей из: 19, 24, 26, 28, 30, 32, 34, 35, 36, 37, 38, 40, 42, 44, 46, 48, 59, 68, 70, 72, 75, 76 77, 78, 80, 82, 168, 180, 182, 184, 186, 188, 189, 190, 191, 192, 193, 195, 197, 199, 201, 203, 223, 225, 227, 229, 231, 232, 234, 236, 238 и 240 аминокислотной последовательности I-OnuI LHE, представленной в SEQ ID NO: 1-5, или соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант HE расщепляет сайт-мишень экзона 5 PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно по меньшей мере 25, более предпочтительно по меньшей мере 35 или даже более предпочтительно по меньшей мере 40 или более аминокислотных замен по меньшей мере в одной положении, выбранном из группы положений, состоящей из положений: 26, 28, 32, 34, 35, 36, 37, 38, 40, 42, 44, 46, 48, 59, 68, 70, 72, 75, 76, 78, 80, 138, 143, 159, 168, 178, 180, 182, 184, 186, 188, 189, 190, 191, 192, 193, 197, 199, 201, 203, 207, 223, 224, 225, 227, 229, 232, 236 и 238 любой из SEQ ID NO: 1-5, или соответствующий биологически активный фрагмент.
В некоторых вариантах реализации вариант HE расщепляет сайт-мишень экзона 5 PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно по меньшей мере 25, более предпочтительно по меньшей мере 35 или еще более предпочтительно по меньшей мере 40 или более, или все из следующих аминокислотных замен: L26Q, R28E, N32V, K34P, S35N, S36I, V37P, G38R, S40K, S40R, E42R, G44R, Q46E, T48D, N59D, V68K, A70Y, S72Q, S72R, N75S, A76Y, S78K, K80R, L138M, T143N, S159P, F168L, E178D, C180H, F182G, N184I, I186A, S188R, K189R, S190P, K191A, L192S, G193P, Q197R, V199R, S201A, S201M, T203G, K207R, Y223R, I224T, K225R, K229I, F232K, D236E и V238E любой из SEQ ID NO: 1-5, или соответствующий биологически активный фрагмент.
В некоторых вариантах реализации вариант HE расщепляет сайт-мишень экзона 5 PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно по меньшей мере 25, более предпочтительно по меньшей мере 35 или еще более предпочтительно по меньшей мере 40 или более, или все из следующих аминокислотных замен: L26Q, R28E, N32V, K34P, S35N, S36I, V37P, G38R, S40K, E42R, G44R, Q46E, T48D, V68K, A70Y, S72Q, N75S, A76Y, S78K, K80R, L138M, T143N, S159P, F168L, C180H, F182G, N184I, I186A, S188R, K189R, S190P, K191A, L192S, G193P, Q197R, V199R, S201A, T203G, K207R, Y223R, K225R, K229I, F232K, D236E и V238E любой из SEQ ID NO: 1-5, или соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант HE расщепляет сайт-мишень экзона 5 PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно, по меньшей мере 25, более предпочтительно, по меньшей мере 35 или даже более предпочтительно, по меньшей мере 40 или более, или все следующих аминокислотных замен: L26Q, R28E, N32V, K34P, S35N, S36I, V37P, G38R, S40R, E42R, G44R, Q46E, T48D, N59D, V68K, A70Y, S72R, N75S, A76Y, K80R, L138M, T143N, S159P, F168L, C180H, F182G, N184I, I186A, S188R, K189R, S190P, K191A, L192S, G193P, Q197R, V199R, S201A, T203G, K207R, Y223R, I224T, K225R, K229I, F232K, D236E и V238E любой из SEQ ID NO: 1-5, или соответствующий биологически активный фрагмент.
В дополнительных вариантах реализации вариант HE расщепляет сайт-мишень экзона 5 PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно по меньшей мере 25, более предпочтительно по меньшей мере 35 или еще более предпочтительно по меньшей мере 40 или более, или все из следующих аминокислотных замен: L26Q, R28E, N32V, K34P, S35N, S36I, V37P, G38R, S40R, E42R, G44R, Q46E, T48D, N59D, V68K, A70Y, S72Q, N75S, A76Y, K80R, L138M, T143N, S159P, F168L, E178D, C180H, F182G, N184I, I186A, S188R, K189R, S190P, K191A, L192S, G193P, Q197R, V199R, S201A, T203G, K207R, Y223R, I224T, K225R, K229I, F232K, D236E и V238E любой из SEQ ID NO: 1-5, или соответствующий биологически активный фрагмент.
В дополнительных вариантах реализации вариант HE расщепляет сайт-мишень экзона 5 PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно, по меньшей мере 25, более предпочтительно, по меньшей мере 35 или даже более предпочтительно, по меньшей мере 40 или более, или все из следующих аминокислотных замен: L26Q, R28E, N32V, K34P, S35N, S36I, V37P, G38R, S40R, E42R, G44R, Q46E, T48D, N59D, V68K, A70Y, S72Q, N75S, A76Y, K80R, L138M, T143N, S159P, F168L, E178D, C180H, F182G, N184I, I186A, S188R, K189R, S190P, K191A, L192S, G193P, Q197R, V199R, S201A, T203G, K207R, Y223R, K225R, F232K, D236E и V238E любой из SEQ ID NO: 1-5, или соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант HE расщепляет сайт-мишень экзона 5 PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно, по меньшей мере 25, более предпочтительно, по меньшей мере 35 или даже более предпочтительно, по меньшей мере 40 или более, или все из следующих аминокислотных замен: L26Q, R28E, N32V, K34P, S35N, S36I, V37P, G38R, S40R, E42R, G44R, Q46E, T48D, N59D, V68K, A70Y, S72Q, N75S, A76Y, K80R, L138M, T143N, S159P, F168L, E178D, C180H, F182G, N184I, I186A, S188R, K189R, S190P, K191A, L192S, G193P, Q197R, V199R, S201M, T203G, Y223R, K225R, F232K, D236E и V238E любой из SEQ ID NO: 1-5, или соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант HE расщепляет сайт-мишень экзона 1 PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно по меньшей мере 25, более предпочтительно по меньшей мере 35 или даже более предпочтительно по меньшей мере 40 или более аминокислотных замен по меньшей мере в одной позиции, выбранной из группы позиций, состоящей из позиций: 26, 28, 30, 32, 34, 35, 36, 37, 38, 40, 42, 44, 46, 48, 68, 70, 72, 75, 76, 78, 80, 100, 132, 138, 143, 155, 159, 178, 180, 184, 186, 189, 190, 191, 192, 193, 195, 201, 203, 207, 223, 225, 227, 232, 236, 238 и 240 любой из SEQ ID NO: 1-5, или соответствующий биологически активный фрагмент.
В некоторых вариантах реализации вариант HE расщепляет сайт-мишень экзона 1 PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно по меньшей мере 25, более предпочтительно по меньшей мере 35 или даже более предпочтительно по меньшей мере 40 или более аминокислотных замен по меньшей мере в одной позиции, выбранной из группы позиций, состоящей из позиций: L26G, R28S, R30L, N32R, K34R, S35G, S36T, V37A, V37G, G38R, S40H, E42R, G44S, Q46A, Q46T, T48V, T48M, V68I, V68S, A70T, A70Y, A70L, S72D, S72N, N75R, N75H, A76Y, S78R, S78T, K80R, K80C, K80E, K80V, T82F, T82Y, I100V, V132A, L138M, T143N, S155G, S159P, E178D, C180S, N184R, I186R, K189N, S190V, K191N, L192A, G193R, Q195R, S201E, T203S, K207R, Y223H, K225Y, K227G, F232R, D236Q, V238R и T240E любой из SEQ ID NO: 1-5, или соответствующий биологически активный фрагмент.
В дополнительных вариантах реализации вариант HE расщепляет сайт-мишень экзона 1 PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно, по меньшей мере 25, более предпочтительно, по меньшей мере 35 или даже более предпочтительно, по меньшей мере 40 или более из, или все из следующих аминокислотных замен: L26G, R28S, R30L, N32R, K34R, S35G, S36T, V37A, G38R, S40H, E42R, G44S, Q46A, T48V, V68I, A70T, S72D, N75R, A76Y, S78R, K80R, I100V, L138M, T143N, S159P, E178D, C180S, N184R, I186R, K189N, S190V, K191N, L192A, G193R, Q195R, S201E, T203S, K207R, Y223H, K225Y, K227G, F232R, D236Q, V238R и T240E любой из SEQ ID NO: 1-5, или соответствующий биологически активный фрагмент.
В некоторых вариантах реализации вариант HE расщепляет сайт-мишень экзона 1 PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно, по меньшей мере 25, более предпочтительно, по меньшей мере 35 или даже более предпочтительно, по меньшей мере 40 или более из, или все из следующих аминокислотных замен: L26G, R28S, R30L, N32R, K34R, S35G, S36T, V37A, V37G, G38R, S40H, E42R, G44S, Q46T, T48V, V68I, A70T, S72D, N75R, A76Y, S78R, K80C, I100V, V132A, L138M, T143N, S155G, S159P, E178D, C180S, N184R, I186R, K189N, S190V, K191N, L192A, G193R, Q195R, S201E, T203S, K207R, Y223H, K225Y, K227G, F232R, D236Q, V238R и T240E любой из SEQ ID NO: 1-5, или соответствующий биологически активный фрагмент.
В некоторых вариантах реализации вариант HE расщепляет сайт-мишень экзона 1 PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно, по меньшей мере 25, более предпочтительно, по меньшей мере 35 или даже более предпочтительно, по меньшей мере 40 или более из, или все из следующих аминокислотных замен: L26G, R28S, R30L, N32R, K34R, S35G, S36T, V37A, G38R, S40H, E42R, G44S, Q46T, T48M, V68I, A70T, S72N, N75H, A76Y, S78T, K80R, I100V, L138M, T143N, S159P, E178D, C180S, N184R, I186R, K189N, S190V, K191N, L192A, G193R, Q195R, S201E, T203S, K207R, Y223H, K225Y, K227G, F232R, D236Q, V238R и T240E любой из SEQ ID NO: 1-5, или соответствующий биологически активный фрагмент.
В некоторых вариантах реализации вариант HE расщепляет сайт-мишень экзона 1 PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно, по меньшей мере 25, более предпочтительно, по меньшей мере 35 или даже более предпочтительно, по меньшей мере 40 или более из, или все из следующих аминокислотных замен: L26G, R28S, R30L, N32R, K34R, S35G, S36T, V37A, G38R, S40H, E42R, G44S, Q46T, T48M, V68S, A70Y, S72N, N75H, A76Y, K80E, T82F, L138M, T143N, S159P, E178D, C180S, N184R, I186R, K189N, S190V, K191N, L192A, G193R, Q195R, S201E, T203S, K207R, Y223H, K225Y, K227G, F232R, D236Q, V238R и T240E любой из SEQ ID NO: 1-5, или соответствующий биологически активный фрагмент.
В некоторых вариантах реализации вариант HE расщепляет сайт-мишень экзона 1 PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно, по меньшей мере 25, более предпочтительно, по меньшей мере 35 или даже более предпочтительно, по меньшей мере 40 или более из, или все из следующих аминокислотных замен: L26G, R28S, R30L, N32R, K34R, S35G, S36T, V37A, G38R, S40H, E42R, G44S, Q46T, T48M, V68S, A70L, S72N, N75H, A76Y, K80V, T82Y, L138M, T143N, S159P, E178D, C180S, N184R, I186R, K189N, S190V, K191N, L192A, G193R, Q195R, S201E, T203S, K207R, Y223H, K225Y, K227G, F232R, D236Q, V238R и T240E любой из SEQ ID NO: 1-5, или соответствующий биологически активный фрагмент.
В некоторых вариантах реализации вариант HE расщепляет сайт-мишень экзона 1 PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно по меньшей мере 25, более предпочтительно по меньшей мере 35 или даже более предпочтительно по меньшей мере 40 или более из, или все из следующих аминокислотных замен: L26G, R28S, R30L, N32R, K34R, S35G, S36T, V37G, G38R, S40H, E42R, G44S, Q46T, T48M, V68S, A70T, S72N, N75H, A76Y, K80V, T82Y, L138M, T143N, S159P, E178D, C180S, N184R, I186R, K189N, S190V, K191N, L192A, G193R, Q195R, S201E, T203S, K207R, Y223H, K225Y, K227G, F232R, D236Q, V238R и T240E любой из SEQ ID NO: 1-5, или соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант HE расщепляет сайт-мишень экзона 2 PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно по меньшей мере 25, более предпочтительно по меньшей мере 35 или еще более предпочтительно по меньшей мере 40 или более аминокислотных замен в по меньшей мере одной позиции, выбранной из группы позиций, состоящей из позиций: 24, 26, 28, 30, 32, 34, 35, 36, 37, 38, 40, 41, 42, 44, 46, 48, 68, 70, 72, 74, 75, 76, 78, 80, 82, 116, 138, 143, 159, 168, 178, 180, 182, 184, 186, 188, 189, 190, 191, 192, 193, 195, 199, 203, 207, 225, 227, 229, 232, 236 и 238 любой из SEQ ID NO: 1-5, или соответствующий биологически активный фрагмент.
В дополнительных вариантах реализации вариант HE расщепляет сайт-мишень экзона 2 PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно по меньшей мере 25, более предпочтительно по меньшей мере 35 или даже более предпочтительно по меньшей мере 40 или более аминокислотных замен по меньшей мере в одной позиции, выбранной из группы позиций, состоящей из позиций: S24C, L26Q, R28Y, R28H, R30S, N32V, N32L, K34N, K34R, S35N, S35T, S36R, V37S, V37T, G38R, G38K, S40R, T41A, E42R, G44S, G44R, Q46E, Q46A, T48E, V68I, A70N, S72I, D74N, N75T, N75R, A76S, A76R, S78R, K80S, T82G, T82R, V116L, L138M, T143N, S159P, F168L, E178D, C180N, F182Y, N184H, I186K, K189G, S190R, K191T, L192T, G193R, Q195Y, V199R, T203A, T203S, K207R, K225N, K225T, K227W, K227S, K229A, K229P, F232R, W234A, W234D, D236E и V238R любой из SEQ ID NO: 1-5, или соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант HE расщепляет сайт-мишень экзона 2 PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно по меньшей мере 25, более предпочтительно по меньшей мере 35 или еще более предпочтительно по меньшей мере 40 или более из, или все из следующих аминокислотных замен: L26Q, R28Y, R30S, N32V, K34N, S35N, S36R, V37S, G38R, S40R, T41A, E42R, G44R, Q46A, T48E, A70N, S72I, N75T, A76S, S78R, K80S, T82G, L138M, T143N, S159P, F168L, E178D, C180N, F182Y, N184H, I186K, K189G, S190R, K191T, L192T, G193R, Q195Y, V199R, T203A, K207R, K225N, K227W, K229A, F232R, W234A, D236E и V238R любой из SEQ ID NO: 1-5, или соответствующий биологически активный фрагмент.
В некоторых вариантах реализации вариант HE расщепляет сайт-мишень экзона 2 PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно по меньшей мере 25, более предпочтительно по меньшей мере 35 или еще более предпочтительно по меньшей мере 40 или более из, или все из следующих аминокислотных замен: S24C, R28H, N32L, K34R, S35T, V37T, G38K, S40R, E42R, G44S, Q46E, T48E, V68I, A70N, S72I, D74N, N75R, A76R, S78R, K80S, T82R, V116L, L138M, T143N, S159P, F168L, E178D, C180N, F182Y, N184H, I186K, K189G, S190R, K191T, L192T, G193R, Q195Y, V199R, T203S, K207R, K225T, K227S, K229P, F232R, W234D, D236E и V238R любой из SEQ ID NO: 1-5, или соответствующий биологически активный фрагмент.
В дополнительных вариантах реализации вариант HE содержит аминокислотную последовательность, которая по меньшей мере на 80%, предпочтительно, по меньшей мере на 85%, более предпочтительно, по меньшей мере на 90% или даже более предпочтительно, по меньшей мере на 95%, идентична аминокислотной последовательности, представленной в любой из SEQ ID NO: 6-14, 60-63 или ее соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант HE содержит аминокислотную последовательность, представленную в SEQ ID NO: 6, или соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант HE содержит аминокислотную последовательность, представленную в SEQ ID NO: 7, или соответствующий биологически активный фрагмент.
В некоторых вариантах реализации вариант HE содержит аминокислотную последовательность, представленную в SEQ ID NO: 8, или соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант HE содержит аминокислотную последовательность, представленную в SEQ ID NO: 9, или соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант HE содержит аминокислотную последовательность, представленную в SEQ ID NO: 10, или соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант HE содержит аминокислотную последовательность, представленную в SEQ ID NO: 11, или соответствующий биологически активный фрагмент.
В некоторых вариантах реализации вариант HE содержит аминокислотную последовательность, представленную в SEQ ID NO: 12, или соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант HE содержит аминокислотную последовательность, представленную в SEQ ID NO: 13, или соответствующий биологически активный фрагмент.
В некоторых вариантах реализации вариант HE содержит аминокислотную последовательность, представленную в SEQ ID NO: 14, или соответствующий биологически активный фрагмент.
В некоторых вариантах реализации вариант HE содержит аминокислотную последовательность, представленную в SEQ ID NO: 60, или соответствующий биологически активный фрагмент.
В некоторых вариантах реализации вариант HE содержит аминокислотную последовательность, представленную в SEQ ID NO: 61, или соответствующий биологически активный фрагмент.
В некоторых вариантах реализации вариант HE содержит аминокислотную последовательность, представленную в SEQ ID NO: 62, или соответствующий биологически активный фрагмент.
В некоторых вариантах реализации вариант HE содержит аминокислотную последовательность, представленную в SEQ ID NO: 63, или соответствующий биологически активный фрагмент.
В частных вариантах реализации полипептид связывает полинуклеотидную последовательность, представленную в SEQ ID NO: 25.
В частных вариантах реализации полипептид связывает полинуклеотидную последовательность, представленную в SEQ ID NO: 30.
В частных вариантах реализации полипептид связывает полинуклеотидную последовательность, представленную в SEQ ID NO: 35.
В дополнительных вариантах реализации полипептид дополнительно содержит ДНК-связывающий домен.
В некоторых вариантах реализации ДНК-связывающий домен выбран из группы, состоящей из: ДНК-связывающего домена TALE и ДНК-связывающего домена «цинковые пальцы».
В некоторых вариантах реализации ДНК-связывающий домен TALE содержит от приблизительно 9,5 областей повторов TALE до приблизительно 15,5 областей повторов TALE.
В дополнительных вариантах реализации ДНК-связывающий домен TALE связывает полинуклеотидную последовательность в гене PD-1.
В частных вариантах реализации ДНК-связывающий домен TALE связывает полинуклеотидную последовательность, представленную в SEQ ID NO: 26.
В некоторых вариантах реализации полипептид связывает и расщепляет полинуклеотидную последовательность, представленную в SEQ ID NO: 27.
В частных вариантах реализации ДНК-связывающий домен TALE связывает полинуклеотидную последовательность, представленную в SEQ ID NO: 31.
В некоторых вариантах реализации полипептид связывает и расщепляет полинуклеотидную последовательность, представленную в SEQ ID NO: 32.
В частных вариантах реализации ДНК-связывающий домен TALE связывает полинуклеотидную последовательность, представленную в SEQ ID NO: 36.
В определенных вариантах реализации полипептид связывает и расщепляет полинуклеотидную последовательность, представленную в SEQ ID NO: 37.
В некоторых вариантах реализации ДНК-связывающий домен «цинковые пальцы» содержит 2, 3, 4, 5, 6, 7 или 8 мотивов «цинковые пальцы».
В дополнительных вариантах реализации полипептид дополнительно содержит пептидный линкер и фермент, изменяющий структуру концов, или соответствующий биологически активный фрагмент.
В частных вариантах реализации полипептид дополнительно содержит вирусный саморасщепляемый пептид 2А и фермент, изменяющий структуру концов, или соответствующий биологически активный фрагмент.
В дополнительных вариантах реализации фермент, изменяющий структуру концов, или соответствующий биологически активный фрагмент обладает 5'-3'-экзонуклеазной, 5'-3' щелочной экзонуклеазной, 3'-5', 5'-flap-экзонуклеазной, геликазной или матрично-независимой ДНК-полимеразной активностью.
В частных вариантах реализации фермент, изменяющий структуру концов, содержит белок Trex2 или соответствующий биологически активный фрагмент.
В некоторых вариантах реализации полипептид содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 15-23 и 64, или соответствующий биологически активный фрагмент.
В дополнительных вариантах реализации полипептид содержит аминокислотную последовательность, представленную в SEQ ID NO: 15, или соответствующий биологически активный фрагмент.
В частных вариантах реализации полипептид содержит аминокислотную последовательность, представленную в SEQ ID NO: 16, или соответствующий биологически активный фрагмент.
В различных вариантах реализации полипептид содержит аминокислотную последовательность, представленную в SEQ ID NO: 17, или соответствующий биологически активный фрагмент.
В частных вариантах реализации полипептид содержит аминокислотную последовательность, представленную в SEQ ID NO: 18, или соответствующий биологически активный фрагмент.
В частных вариантах реализации полипептид содержит аминокислотную последовательность, представленную в SEQ ID NO: 19, или соответствующий биологически активный фрагмент.
В некоторых вариантах реализации полипептид содержит аминокислотную последовательность, представленную в SEQ ID NO: 20, или соответствующий биологически активный фрагмент.
В некоторых вариантах реализации полипептид содержит аминокислотную последовательность, представленную в SEQ ID NO: 21, или соответствующий биологически активный фрагмент.
В дополнительных вариантах реализации полипептид содержит аминокислотную последовательность, представленную в SEQ ID NO: 22, или соответствующий биологически активный фрагмент.
В дополнительных вариантах реализации полипептид содержит аминокислотную последовательность, представленную в SEQ ID NO: 23, или соответствующий биологически активный фрагмент.
В дополнительных вариантах реализации полипептид содержит аминокислотную последовательность, представленную в SEQ ID NO: 64, или соответствующий биологически активный фрагмент.
В дополнительных вариантах реализации полипептид, рассматриваемый в настоящей заявке, содержит аминокислотную последовательность, представленную в SEQ ID NO: 24, или соответствующий биологически активный фрагмент.
В других вариантах реализации полипептид расщепляет ген PD-1 человека на полинуклеотидной последовательности, представленной в SEQ ID NO: 25, 27, 30, 32, 35 или 37.
В различных вариантах реализации настоящее изобретение, в частности, предусматривает полинуклеотид, кодирующий полипептид, рассматриваемый в настоящей заявке.
В частных вариантах реализации настоящее изобретение, в частности, предусматривает мРНК, кодирующую полипептид, рассматриваемый в настоящей заявке.
В частных вариантах реализации мРНК содержит последовательность, представленную в SEQ ID NO: 40-42 и 65-68.
В различных вариантах реализации настоящее изобретение предусматривает, в частности, кДНК, кодирующую полипептид, рассматриваемый в настоящей заявке.
В определенных вариантах реализации настоящее изобретение, в частности,предусматривает вектор, содержащий полинуклеотид, кодирующий полипептид, рассматриваемый в настоящей заявке.
В различных вариантах реализации настоящее изобретение, в частности, предусматривает клетку, содержащую полипептид, рассматриваемый в настоящей заявке.
В некоторых вариантах реализации настоящее изобретение, в частности, предусматривает клетку, содержащую полинуклеотид, кодирующий полипептид, рассматриваемый в настоящей заявке.
В различных вариантах реализации настоящее изобретение, в частности, предусматривает клетку, содержащую вектор, рассматриваемый в настоящей заявке.
В дополнительных вариантах настоящее изобретение, в частности, предусматривает клетку, содержащую одну или более модификаций генома, введенных полипептидом, рассматриваемым в настоящей заявке.
В частных вариантах реализации клетка содержит полинуклеотид, кодирующий один или более группы, состоящей из: энхансера иммунопотенции, демпфера иммуносупрессивного сигнала или сконструированного рецептора антигена.
В некоторых вариантах реализации полинуклеотид дополнительно содержит промотор РНК-полимеразы II, функционально связанный с полинуклеотидом, кодирующим энхансер иммунопотенции, демпфер иммуносупрессивного сигнала или сконструированный рецептор антигена.
В частных вариантах реализации промотор РНК-полимеразы II выбран из группы, состоящей из короткого промотора EF1α, длинного промотора EF1α, локуса ROSA 26 человека, промотора убиквитина C (UBC), промотора фосфоглицераткиназы-1 (PGK), энхансера цитомегаловируса/промотора β-актина курицы (CAG), промотора β-актина и энхансера вируса миелопролиферативной саркомы с делецией опосредующего отрицательную регуляцию участка, с заменой на праймер-связывающий сайт из dl587rev (MND).
В некоторых вариантах реализации полинуклеотид дополнительно кодирует один или более саморасщепляемых вирусных пептидов, функционально связанных, рассеянных между и/или фланкирующих энхансер иммунопотенции, демпфер иммуносупрессивного сигнала или сконструированный рецептор антигена.
В некоторых вариантах реализации саморасщепляемый вирусный пептид представляет собой пептид 2А.
В определенных вариантах реализации полинуклеотид дополнительно содержит гетерологичный сигнал полиаденилирования.
В некоторых вариантах реализации демпфер иммуносупрессивного сигнала включает ферментативную функцию, которая противодействует иммуносупрессорному фактору.
В некоторых вариантах реализации демпфер иммуносупрессивного сигнала включает кинурениназную активность.
В частных вариантах реализации демпфер иммуносупрессивного сигнала содержит: экзодомен, который связывает иммуносупрессивный фактор, причем, необязательно, указанный экзодомен представляет собой антитело или его антигенсвязывающий фрагмент; экзодомен, который связывает иммуносупрессивный фактор и трансмембранный домен; или экзодомен, который связывает иммуносупрессивный фактор, трансмембранный домен и модифицированный эндодомен, который не способен трансдуцировать иммуносупрессивные сигналы в клетку.
В некоторых вариантах реализации демпфер иммуносупрессивного сигнала представляет собой доминантный отрицательный рецептор TGFβRII.
В некоторых вариантах реализации энхансер иммунопотенции выбран из группы, состоящей из: молекулы биспецифического Т-клеточного активатора (BiTE), иммуноопотенцирующего фактора и флип-рецептора.
В частных вариантах реализации иммунопотенцирующий фактор выбран из группы, состоящей из цитокина, хемокина, цитотоксина, рецептора цитокина и их вариантов.
В частных вариантах реализации рецептор цитокина выбран из группы, состоящей из рецептора IL-2, рецептора IL-7, рецептора IL-12, рецептора IL-15, рецептора IL-18 и рецептора IL-21.
В предпочтительном варианте реализации клетка содержит полинуклеотид, кодирующий рецептор цитокина, выбранный из группы, состоящей из рецептора IL-2, рецептора IL-7, рецептора IL-12, рецептора IL-15, рецептора IL-18, и рецептора IL-21, функционально связанный с эндогенным промотором PD-1.
В другом предпочтительном варианте реализации клетка содержит полинуклеотид, кодирующий рецептор цитокина IL-12, функционально связанный с эндогенным промотором PD-1.
В частных вариантах реализации цитокин выбран из группы, состоящей из IL-2, IL-7, IL-12, IL-15, IL-18 и IL-21.
В предпочтительном варианте реализации клетка содержит полинуклеотид, кодирующий цитокин, выбранный из группы, состоящей из IL-2, IL-7, IL-12, IL-15, IL-18 и IL-21, функционально связанных с эндогенным промотором PD-1.
В другом предпочтительном варианте реализации клетка содержит полинуклеотид, кодирующий IL-12, функционально связанный с эндогенным промотором PD-1.
В дополнительных вариантах реализации флип-рецептор содержит экзодомен PD-1 и трансмембранный домен; и эндодомен из CD28, CD134, CD137, CD278 и/или CD3ζ, слитые в рамке с C-концевым концом трансмембранного домена PD-1.
В определенных вариантах реализации, флип-рецептор содержит экзодомен PD-1; трансмембранный домен, выделенный из полипептида CD3, CD4, CD8α, CD28, CD134 или CD137; и эндодомен из CD28, CD134, CD137, CD278 и/или CD3ζ, слитые в рамке с C-концевым концом экзодомена PD-1.
В частных вариантах реализации флип-рецептор содержит экзодомен PD-1; а также трансмембранный домен и эндодомен, выделенные из полипептида CD3, CD4, CD8α, CD28, CD134 или CD137, слитые в рамке с C-концевым концом экзодомена PD-1.
В дополнительных вариантах реализации сконструированный рецептор антигена выбран из группы, состоящей из: сконструированного TCR, CAR, Daric или дзетакина.
В частных вариантах сконструированный рецептор не встроен в ген PD-1.
В некоторых вариантах реализации полинуклеотид, кодирующий один или более из: энхансера иммунопотенции, демпфера иммуносупрессивного сигнала или сконструированного рецептора антигена, встроен в ген PD-1.
В других вариантах реализации матрица донорной репарации, содержащая полинуклеотид, кодирующий один или более из: энхансера иммунопотенции, демпфера иммуносупрессивного сигнала или сконструированный рецептор антигена, встроен в ген PD-1 в сайте двухцепочечного разрыва ДНК, введенном посредством полипептида, рассматриваемого в настоящей заявке.
В частных вариантах реализации донорная матрица репарации, содержащая полинуклеотид, кодирующий цитокин, выбранный из группы, состоящей из IL-2, IL-7, IL-12, IL-15, IL-18 и IL-21, встроена в PD-1 ген в сайте двухцепочечного разрыва ДНК, введенном полипептидом, рассматриваемым в настоящей заявке. В предпочтительных вариантах реализации цитокин встроен в ген PD-1 в функциональной связи с эндогенным промотором PD-1.
В частных вариантах реализации донорная матрица репарации, содержащая полинуклеотид, кодирующий цитокин IL-12, встроена в ген PD-1 в сайте двухцепочечного разрыва ДНК, введенном полипептидом, рассматриваемым в настоящей заявке. В предпочтительных вариантах реализации цитокин IL-12 встроен в ген PD-1 в функциональной связи с эндогенным промотором PD-1.
В частных вариантах реализации донорная матрица репарации содержит полинуклеотид, кодирующий рецептор цитокина, выбранный из группы, состоящей из рецептора IL-2, рецептора IL-7, рецептора IL-12, рецептора IL-15, рецептора IL-18 и рецептора IL-21, встроена в ген PD-1 в сайте двухцепочечного разрыва ДНК, введенном полипептидом, рассматриваемым в настоящей заявке. В предпочтительных вариантах цитокиновый рецептор встроен в ген PD-1 в функциональной связи с эндогенным промотором PD-1.
В частных вариантах реализации донорная матрица репарации, содержащая полинуклеотид, кодирующий рецептор цитокина IL-12, встроена в ген PD-1 в сайте двухцепочечного разрыва ДНК, введенном полипептидом, рассматриваемым в настоящей заявке. В предпочтительных вариантах реализации рецептор цитокинов IL-12 встроен в ген PD-1 в функциональной связи с эндогенным промотором PD-1.
В некоторых вариантах реализации клетка представляет собой гемопоэтическую клетку.
В дополнительных вариантах реализации клетка представляет собой Т-клетку.
В частных вариантах реализации клетка представляет собой клетку CD3+, CD4+и/или CD8+.
В частных вариантах реализации клетка представляет собой иммунную эффекторную клетку.
В других вариантах реализации клетка представляет собой цитотоксический Т-лимфоцит (ЦТЛ), инфильтрирующий опухоль лимфоцит (TIL) или хелперную Т-клетку.
В некоторых вариантах реализации клетка представляет собой природный киллер (NK) или T-клетку природный киллер (NKT).
В частных вариантах реализации источником клеток являются мононуклеарные клетки периферической крови, костный мозг, ткань лимфатических узлов, пуповинная кровь, ткань тимуса, ткань из места инфекции, асцит, плевральный выпот, ткань селезенки или опухоль.
В частных вариантах реализации настоящее изобретение предусматривает, в частности, совокупность клеток, содержащих одну или более клеток, рассматриваемых в настоящей заявке.
В различных вариантах реализации настоящее изобретение предусматривает, в частности, композицию, содержащую одну или более клеток, рассматриваемых в настоящей заявке.
В определенных вариантах реализации настоящее изобретение предусматривает, в частности, композицию, содержащую одну или более клеток, рассматриваемых в настоящей заявке, и физиологически приемлемый носитель.
В различных вариантах реализации настоящее изобретение предусматривает, в частности, способ редактирования гена PD-1 человека в клетке, включающий введение полинуклеотида, кодирующего полипептид, рассматриваемый в настоящей заявке, в клетку, при котором экспрессия данного полипептида создает разрыв двойной цепи в сайте-мишени в гене PD-1 человека.
В некоторых вариантах реализации настоящее изобретение, в частности, предусматривает способ редактирования гена PD-1 человека в клетке, включающий: введение полинуклеотида, кодирующего полипептид, рассматриваемый в настоящей заявке, в клетку, при котором экспрессия данного полипептида создает двухцепочечный разрыв в сайте-мишени в гене PD-1 человека, причем указанный разрыв репарируется негомологичным соединением концов (NHEJ).
В некоторых вариантах реализации настоящее изобретение, в частности, предусматривает способ редактирования гена PD-1 человека в клетке, включающий введение полинуклеотида, кодирующего полипептид, рассматриваемый в настоящей заявке, и донорной матрицы репарации в клетку, при котором экспрессия полипептида создает двухцепочечный разрыв в сайте-мишени в гене PD-1 человека, и донорная матрица репарации включается в ген PD-1 человека посредством гомологичной репарации (HDR) в сайте двухцепочечного разрыва (DSB).
В других вариантах реализации клетка представляет собой гемопоэтическую клетку.
В частных вариантах реализации клетка представляет собой Т-клетку.
В частных вариантах реализации клетка представляет собой клетку CD3+, CD4+и/или CD8+.
В определенных вариантах реализации клетка представляет собой иммунную эффекторную клетку.
В некоторых вариантах реализации клетка представляет собой цитотоксический Т-лимфоцит (ЦТЛ), инфильтрирующий опухоль лимфоцит (TIL) или хелперную Т-клетку.
В частных вариантах реализации клетка представляет собой клетку природный киллер (NK) или T-клетку природный киллер (NKT).
В некоторых вариантах реализации источником клеток являются мононуклеарные клетки периферической крови, костный мозг, ткань лимфатических узлов, пуповинная кровь, ткань тимуса, ткань из места инфекции, асцит, плевральный выпот, ткань селезенки или опухоль.
В частных вариантах реализации полинуклеотид, кодирующий полипептид, представляет собой мРНК.
В дополнительных вариантах реализации в клетку вводят полинуклеотид, кодирующий 3'-5' экзонуклеазу.
В некоторых вариантах реализации в клетку вводят полинуклеотид, кодирующий Trex2 или соответствующий биологически активный фрагмент.
В других вариантах реализации донорная матрица репарации кодирует ген PD-1 или его часть, содержащую одну или более мутаций по сравнению с геном PD-1 дикого типа.
В частных вариантах реализации донорная матрица репарации кодирует один или более из: энхансера иммунопотенции, демпфера иммуносупрессивного сигнала или сконструированного рецептора антигена.
В дополнительных вариантах реализации донорная матрица репарации дополнительно содержит промотор РНК-полимеразы II, функционально связанный с энхансером иммунопотенции, демпфером иммуносупрессивного сигнала или сконструированным рецептором антигена.
В других вариантах промотор РНК-полимеразы II выбран из группы, состоящей из короткого промотора EF1α, длинного промотора EF1α, локуса ROSA 26 человека, промотора убиквитина C (UBC), промотора фосфоглицераткиназы-1 (PGK), энхансера цитомегаловируса/промотора β-актина курицы (CAG), промотора β-актина и энхансера вируса миелопролиферативной саркомы с делецией опосредующего отрицательную регуляцию участка, с заменой на праймер-связывающий сайт из dl587rev (MND).
В определенных вариантах реализации донорная матрица репарации дополнительно кодирует один или более саморасщепляемых вирусных пептидов, функционально связанных, рассеянных между и/или фланкирующих энхансер иммунопотенции, демпфер иммуносупрессивного сигнала или сконструированный рецептор антигена.
В дополнительных вариантах реализации саморасщепляемый вирусный пептид представляет собой пептид 2А.
В некоторых вариантах реализации донорная матрица репарации дополнительно содержит гетерологичный сигнал полиаденилирования.
В некоторых вариантах реализации демпфер иммуносупрессивного сигнала включает ферментативную функцию, которая противодействует иммуносупрессорному фактору.
В других вариантах реализации демпфер иммуносупрессивного сигнала включает кинурениназную активность.
В частных вариантах реализации демпфер иммуносупрессивного сигнала содержит: экзодомен, который связывает иммуносупрессивный фактор, причем, необязательно, указанный экзодомен представляет собой антитело или его антигенсвязывающий фрагмент; экзодомен, который связывает иммуносупрессивный фактор и трансмембранный домен; или экзодомен, который связывает иммуносупрессивный фактор, трансмембранный домен и модифицированный эндодомен, который не способен трансдуцировать иммуносупрессивные сигналы в клетку.
В дополнительных вариантах реализации демпфер иммуносупрессивного сигнала представляет собой доминантный отрицательный рецептор TGFβRII.
В некоторых вариантах реализации энхансер иммунопотенции выбран из группы, состоящей из: молекулы биспецифического Т-клеточного активатора (BiTE), иммуноопотенцирующего фактора и флип-рецептора.
В других вариантах реализации иммунопотенцирующий фактор выбран из группы, состоящей из цитокина, хемокина, цитотоксина, рецептора цитокина и их вариантов.
В частных вариантах реализации иммунопотенцирующий фактор выбран из группы, состоящей из цитокина, хемокина, цитотоксина, рецептора цитокина и их вариантов.
В частных вариантах реализации рецептор цитокина выбран из группы, состоящей из рецептора IL-2, рецептора IL-7, рецептора IL-12, рецептора IL-15, рецептора IL-18 и рецептора IL-21.
В предпочтительном варианте реализации клетка содержит полинуклеотид, кодирующий рецептор цитокина, выбранный из группы, состоящей из рецептора IL-2, рецептора IL-7, рецептора IL-12, рецептора IL-15, рецептора IL-18, и рецептора IL-21, функционально связанный с эндогенным промотором PD-1.
В другом предпочтительном варианте реализации клетка содержит полинуклеотид, кодирующий рецептор цитокина IL-12, функционально связанный с эндогенным промотором PD-1.
В частных вариантах реализации цитокин выбран из группы, состоящей из IL-2, IL-7, IL-12, IL-15, IL-18 и IL-21.
В предпочтительном варианте реализации клетка содержит полинуклеотид, кодирующий цитокин, выбранный из группы, состоящей из IL-2, IL-7, IL-12, IL-15, IL-18 и IL-21, функционально связанных с эндогенным промотором PD-1.
В другом предпочтительном варианте реализации клетка содержит полинуклеотид, кодирующий IL-12, функционально связанный с эндогенным промотором PD-1.
В частных вариантах реализации флип-рецептор содержит экзодомен PD-1 и трансмембранный домен; и эндодомен из CD28, CD134, CD137, CD278 и/или CD3ζ, слитые в рамке с C-концевым концом трансмембранного домена PD-1.
В дополнительных вариантах реализации, флип-рецептор содержит экзодомен PD-1; трансмембранный домен, выделенный из полипептида CD3, CD4, CD8α, CD28, CD134 или CD137; и эндодомен из CD28, CD134, CD137, CD278 и/или CD3ζ, слитые в рамке с C-концевым концом экзодомена PD-1.
В других вариантах реализации, флип-рецептор содержит экзодомен PD-1; а также трансмембранный домен и эндодомен, выделенные из полипептида CD3, CD4, CD8α, CD28, CD134 или CD137, слитые в рамке с C-концевым концом экзодомена PD-1.
В дополнительных вариантах реализации сконструированный рецептор антигена выбран из группы, состоящей из: сконструированного TCR, CAR, Daric или дзетакина.
В дополнительных вариантах реализации, донорная матрица репарации содержит 5'-гомологичное плечо, гомологичное 5'-последовательности гена PD-1 человека DSB, и 3'- гомологичное плечо, гомологичное 3'-последовательности гена PD-1 человека DSB.
В частных вариантах реализации длины 5'- и 3'-гомологичных плеч выбраны независимо от приблизительно 100 до приблизительно 2500 п.н.
В некоторых вариантах реализации длины 5'- и 3'-гомологичных плеч независимо выбраны от приблизительно 600 до приблизительно 1500 п.н.
В некоторых вариантах 5'-гомологичное плечо составляет приблизительно 1500 п.н., а 3'- гомологичное плечо составляет приблизительно 1000 п.н.
В некоторых вариантах реализации 5'-гомологичное плечо составляет приблизительно 600 п.н., а 3'-гомологичное плечо составляет приблизительно 600 п.н.
В частных вариантах реализации для введения донорной матрицы репарации в клетку используют вирусный вектор.
В дополнительных вариантах реализации вирусный вектор представляет собой рекомбинантный адено-ассоциированный вирусный вектор (rAAV) или ретровирус.
В других вариантах реализации, указанный rAAV имеет один или более ITR из AAV2.
В некоторых вариантах реализации rAAV имеет серотип, выбранный из группы, состоящей из: AAV1, AAV2, AAV3, AAV4, AAV5, AAV6, AAV7, AAV8, AAV9 и AAV10.
В дополнительных вариантах реализации, rAAV имеет серотип AAV2 или AAV6.
В некоторых вариантах реализации ретровирус представляет собой лентивирус.
В частных вариантах реализации лентивирус является дефицитным по интегразе лентивирусом (IDLV).
В различных вариантах реализации настоящее изобретение, в частности, предусматривает способ лечения, предотвращения или ослабления по меньшей мере одного симптома рака, инфекционного заболевания, аутоиммунного заболевания, воспалительного заболевания и иммунодефицита или состояния, ассоциированного с ними, включающий введение субъекту эффективного количества композиции, рассматриваемой в настоящей заявке.
В различных вариантах реализации настоящее изобретение, в частности, предусматривает способ лечения солидного рака, включающий введение субъекту эффективного количества композиции, рассматриваемой в настоящей заявке.
В других вариантах реализации, солидный рак включает рак печени, рак поджелудочной железы, рак легких, рак молочной железы, рак яичников, рак предстательной железы, рак яичка, рак мочевого пузыря, рак мозга, саркому, рак головы и шеи, рак кости, рак щитовидной железы, рак почки или рак кожи.
В различных вариантах реализации настоящее изобретение, в частности, предусматривает способ лечения гематологического злокачественного заболевания, включающий введение субъекту эффективного количества композиции, рассматриваемой в настоящей заявке.
В дополнительных вариантах реализации, указанное гематологическое злокачественное заболевание представляет собой лейкоз, лимфому или множественную миелому.
Краткое описание нескольких видов чертежей
Фигура 1A. Схема, иллюстрирующая положения домена IgV и мотивов ITIM и ITSM PD-1 относительно положения сайта-мишени megaTAL в экзоне.
Фигура 1B. Ген PD-1 и последовательность сайта-мишень в мотиве ITSM, кодируемого экзоном 5 (SEQ ID NO: 106 - 108), выделен мотив центральный-4 HE, расположенный в кодоне для остатка фосфотирозина ITSM в положении 248.
Фигура 2. I-OnuI был перепрограммирован посредством конструирования NTD (N-концевой домен) и CTD (С-концевой домен) для химерных «полу-сайтов» (SEQ ID NO: 109 и 110) с помощью двух раундов сортировки с последующим слиянием перепрограммированных доменов (SEQ ID NO: 111) и скрининга на полный экзон PD-1 5 сайт-мишень, чтобы изолировать полностью перепрограммированное HE.
Фигура 3A. Первоначальный скрининг варианта экзона 5 гена PD-1 на активность в анализе по хромосомному репортеру.
Фигура 3B. Вариант HE PD-1 (PD-1.ITSM.ex5_RD1_CV3-08) обладал умеренными свойствами аффинности связывания ДНК при измерении путем равновесного титрования субстрата.
Фигура 4. Вторичный скрининг HE PD-1 на активность в анализе по хромосомному репортеру после выделения улучшенных вариантов из сортировки потока на основе дисплея случайным образом мутагенизированной библиотеки вариантов PD-1.ITSM.ex5_RD1_CV3-08, выполненной при более строгих условиях расщеплении и аффинности для выделения вариантов с улучшенной активностью.
Фигура 5. Результаты анализа бисульфитного секвенирования экзона 5 PD-1 (SEQ ID NO: 112) в активированных первичных Т-клетках человека для определения статуса метилирования мотивов CpG (SEQ ID NO: 113), присутствующих в экзоне 5 HE PD-1.
Фигура 6. Результаты анализа аффинности связывания и расщепления ДНК для варианта HE PD-1.ITSM.ex5_RD5_CV23MK по сравнению с частично и полностью метилированными субстратами экзона 5 PD-1.
Фигура 7A. Совместная доставка megaTAL PD-1.ITSM.ex5_RD5_CV23MK в T-клетки вместе с TREX2 редактирует локус-мишень на уровне приблизительно 60%, согласно измерениям анализом TIDE.
Фигура 7B. Распределение инделов по консенсусному мотиву PD-1 ITSM при расщеплении megaTAL PD-1.ITSM.ex5_RD5_CV23MK в присутствии Trex2.
Фигура 8. Результаты профилирования центральной-4 специфичности экзона 5 HE PD-1.
Фигура 9A. Схема, иллюстрирующая положения доменов IgV, ITIM и ITSM PD-1 относительно положения экзонов 1, 2 и 5.
Фигура 9B. Ген PD-1 и местоположение сайта-мишени в экзонах 1 (SEQ ID NO: 114 - 116), 2 (SEQ ID NO: 117-119) и 5 (SEQ ID NO: 106-108).
Фигура 10. Результаты анализов хромосомных репортеров после сортировки потока на основе дисплея в отношении каталитической активности для первоначально перепрограммированного I-OnuI, нацеленного на экзон 1 PD-1 (PD-1.ile3.exon1_RD1_B1, верхняя панель); обогащение PD-1.ile3.exon1_RD1_B1 путем мутагенизации и скрининга в более жестких каталитических условиях для выявления мутаций, которые способствуют расщеплению мишени, для выявления более активных вариантов (PD-1.ile3.exon1_RD2_B1H8 и PD-1.ile3.exon1_RD2_B1G2, средние панели); и первоначально перепрограммированного I-OnuI, нацеленного на экзон 2 PD-1 (PD-1.IgV.exon2_RD1_G5, нижняя панель). Результаты показаны для нуклеазы в присутствии Trex2 (левые панели) и отформатированы в виде megaTAL (правые панели).
Фигура 11A. Величины аффинности связывания ДНК для вариантов PD-1.ile3.exon1_RD2_B1H8 и PD-1.IgV.exon2_RD1_G5 при измерении путем равновесного титрования субстрата с применением их соответствующих последовательностей-мишеней.
Фигура 11B. Сравнение активности расщепления ДНК среди обогащенных по специфичности нуклеаз PD-1.ile3.exon1 (RD2_B1G2, RD3_B1G2C4, RD3_B1G2C11, RD3_B1G2C5) в отношении 64 ДНК-мишеней, с вариациями в парах оснований -8, -7 и -6. Теплокарта представляет коэфициент не-расщепления: медианные значения расщепления получены из точечных графиков.
Фигура 12. Результаты анализа бисульфитного секвенирования экзона 1 (SEQ ID NO: 120) и экзона 2 (SEQ ID NO: 122) PD-1 в активированных первичных Т-клетках человека (левые панели), демонстрирующие, что мотив CpG экзона 1 PD-1 (SEQ ID NO: 121) остается неметилированным, в то время как мотивы CpG экзона 2 PD-1 (SEQ ID NO: 123) метилированы. Фигура 12 также показывает, что PD-1.IgV.exon2_RD1_G5 эффективно расщеплял и неметилированный, и метилированный сайт-мишень (правая верхняя и правая нижняя панели, соответственно).
Фигура 13A. Поверхностная экспрессия PD-1 в CAR Т-клетках, электропорированных пустым носителем, CFP, megaTALCCR5, megaTALPD-1.ITSM.ex5_RD5_CV23MK или megaTAL PD-1.ile3.exon1_RD2_B1H8, а затем стимулированных пустым носителем или форбол-12-миристат 13-ацетатом (PMA)/иономицином (P/I).
Фигура 13B. Поверхностная экспрессия PD-1 в Т-клетках, электропорированных усовершенствованными версиями megaTAL PD-1.ile3.exon1 (RD2_B1G2, RD3_ B1G2C4, RD3_B1G2C11, RD3_B1G2C5)
Фигура 14. Одновременная доставка megaTAL PD-1.ITSM.ex5_RD5_CV23MK и megaTAL PD-1.ile3.exon1_RD2_B1H8 с Trex2 или без него значительно снижает экспрессию PD-1 на клеточной поверхности.
Фигура 15A. Анти-BCMA CAR T-клетки, электропорированные мРНК, кодирующей megaTAL PD-1.ITSM.ex5_RD5_CV23MK или PD-1.ile3.exon1_RD2_B1H8, показывают пониженное PD-L1-опосредованное подавление цитокинов по сравнению с анти-BCMA CAR T-клетками, электропорированными пустым носителем или мРНК, кодирующей megaTAL CFP или CCR5.
Фигура 15B. Анти-BCMA CAR T-клетки, электропорированные мРНК, кодирующей megaTAL PD-1.ITSM.ex5_RD5_CV23MK или PD-1.ile3.exon1_RD2_B1H8, показывают пониженное PD-L1-опосредованное подавление цитокинов по сравнению с анти-BCMA CAR T-клетками, электропорированными пустым носителем или мРНК кодирующей каталитически неактивный TCRα- megaTAL (необработанные). Добавление антитела PD-1 к культурам отменяет супрессию цитокинов в анти-BCMA CAR T-клетках, электропорированных пустым носителем, или мРНК, кодирующей каталитически неактивный megaTAL TCRα.
Фигура 16A. Стратегия введения различных кассет экспрессий (GFP, верхняя панель; анти-CD19 CAR, средняя панель и анти-BCMA CAR, нижняя панель) в экзон 1 PD-1 путем гомологичной рекомбинации.
Фигура 16B. Репрезентативные результаты анализов проточной цитометрии для определения долговременной экспрессии хромосомно интегрированных кассет в Т-клетках, обработанных megaTAL PD-1.ile3.exon1_RD2_B1H8 и rAAV-нацеленными векторами, содержащими кассету экспрессии GFP, анти-CD19 CAR или анти-BCMA CAR.
Фигура 17. Стратегия введения репортерного гена mCherry в стартовый кодон PD-1 в экзоне 1 и анализ с помощью проточной цитометрии экспрессии mCherry в Т-клетках, электропорированных пустым носителем или megaTAL PD-1.ile3.exon1_RD2_B1H8 и трансдуцированных пустым носителем или нацеленным на rAAV вектором, кодирующим mCherry как в присутствии, так и в отсутствие 24 часовой обработки PMA/иономицином.
Фигура 18. Стратегия введения кассеты экспрессии MND-промотор-BFP в мотив ITSM в экзоне 5 PD-1 и проточная цитометрия для анализа экспрессии BFP в Т-клетках, электропорированных пустым носителем или PD-1.ITSM.ex5_RD5_CV23MK и трансдуцированных пустым носителем или нацеленным на rAAV вектором, содержащим кассету экспрессии pMND-BFP.
Фигура 19. Стратегия введения кассеты экспрессии MND-промотор-BFP в мотив ITSM в экзоне 5 PD-1 и анализ методом проточной цитометрии для экспрессии PD-1 и BFP в Т-клетках, электропорированных пустым носителем или PD-1.ITSM.ex5_RD5_CV23MK megaTAL и трансдуцированных пустым носителем или нацеленным на rAAV вектором, содержащим кассету экспрессии pMND-BFP.
Фигура 20. Стратегия введения кассеты экспрессии MND-промотор-BFP в мотив ITSM в экзоне 5 PD-1, когда гомологичные плечи содержат одиночные нуклеотидные полиморфизмы (SNP) (верхняя панель). На нижней панели показан анализ проточной цитометрии Т-клеток, электропорированных megaTAL PD-1.ITSM.ex5_RD5_CV23MK и трансдуцированных нацеленным на rAAV вектором, содержащим кассету экспрессии pMND-BFP с гомологичными плечами дикого типа, причем 5'-гомологичное плечо содержит SNP, или 3'-гомологичное плечо содержит SNP.
Фигура 21. Стратегия введения кассеты экспрессии MND-промотор-PD-1.CD28 флип-рецептор в мотив ITSM в экзоне 5 PD-1 и анализ с помощью проточной цитометрии экспрессии PD-1 и BFP в Т-клетках, электропорированных пустым носителем или megaTAL PD- 1.ITSM.ex5_RD5_CV23MK и трансдуцированных пустым носителем или нацеленным на rAAV вектором, содержащим кассету экспрессии pMND-PD-1.CD28- флип-рецептор.
Фигура 22. Стратегия создания крупномасштабной делеции в гене PD-1 путем доставки кассеты megaTAL PD-1.ITSM.ex5_RD5_CV23MK и rAAV, содержащей гомологичные плечи на расстоянии более т.п.н. в направлении против хода транскрипции от экзона 5 сайта-мишени.
Фигура 23. Стратегия встраивания цитокина в ген PD-1, когда экспрессия находится под контролем эндогенного промотора PD-1 (верхняя панель). После 24 часов обработки PMA/иономицином анти-BCMA CAR T-клетки, электропорированные мРНК megaTAL PD-1 и трансдуцированные rAAV, кодирующим IL-12, показали пониженную экспрессию PD-1 по сравнению с клетками, обработанными контролем (нижняя панель).
Фигура 24A. После 24-часовой обработки PMA/иономицином анти-BCMA CAR T-клетки, электропорированные мРНК megaTAL PD-1 и трансдуцированные rAAV, кодирующим IL-12, показали повышенную выработку IL-12 по сравнению с клетками, обработанными контролем (левая панель). Анти-BCMA CAR T-клетки, электропорированные мРНК PD-1 и трансдуцированные rAAV, кодирующим IL-12, и культивируемые в присутствии клеток-мишеней K562-BCMA, показали повышенную выработку IL-12 по сравнению с клетками, обработанными контролем (правая панель).
Фигура 24B. Результаты анализа серийной стимуляции. Клетки K52-BCMA и анти-BCMA CAR T-клетки смешивали, культивировали в течение 7 дней и добавляли дополнительные клетки-мишени K562-BCMA для имитации повторной стимуляции. После рестимуляции анти-BCMA CAR T-клетки, обработанные рекомбинантным IL-12 или обработанные как megaTAL PD-1, так и матрицей HDR IL-12, показали повышенную выработку IFNγ и цитотоксичность по сравнению с клетками, обработанными контролем.
КРАТКОЕ ОПИСАНИЕ ИДЕНТИФИКАТОРОВ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
SEQ ID NO: 1 - аминокислотная последовательность хоминг-эндонуклеазы I-OnuI дикого типа LAGLIDADG (LHE).
SEQ ID NO: 2 - аминокислотная последовательность LHE I-OnuI дикого типа.
SEQ ID NO: 3 - аминокислотная последовательность биологически активного фрагмента LHE I-OnuI дикого типа.
SEQ ID NO: 4 - аминокислотная последовательность биологически активного фрагмента LHE I-OnuI дикого типа.
SEQ ID NO: 5 - аминокислотная последовательность биологически активного фрагмента LHE I-OnuI дикого типа.
SEQ ID NO: 6 - аминокислотная последовательность варианта LHE I-OnuI, перепрограммированного для связывания и расщепления сайта-мишени в гене PD-1 человека.
SEQ ID NO: 7 - аминокислотная последовательность варианта LHE I-OnuI, перепрограммированного для связывания и расщепления сайта-мишени в гене PD-1 человека.
SEQ ID NO: 8 - аминокислотная последовательность варианта LHE I-OnuI, перепрограммированного для связывания и расщепления сайта-мишени в гене PD-1 человека.
SEQ ID NO: 9 - аминокислотная последовательность варианта LHE I-OnuI, перепрограммированного для связывания и расщепления сайта-мишени в гене PD-1 человека.
SEQ ID NO: 10 - аминокислотная последовательность варианта LHE I-OnuI, перепрограммированного для связывания и расщепления сайта-мишени в гене PD-1 человека.
SEQ ID NO: 11 - аминокислотная последовательность варианта LHE I-OnuI, перепрограммированного для связывания и расщепления сайта-мишени в гене PD-1 человека.
SEQ ID NO: 12 - аминокислотная последовательность варианта LHE I-OnuI, перепрограммированного для связывания и расщепления сайта-мишени в гене PD-1 человека.
SEQ ID NO: 13 - аминокислотная последовательность варианта LHE I-OnuI, перепрограммированного для связывания и расщепления сайта-мишени в гене PD-1 человека.
SEQ ID NO: 14 - аминокислотная последовательность варианта LHE I-OnuI, перепрограммированного для связывания и расщепления сайта-мишени в гене PD-1 человека.
SEQ ID NO: 15 - аминокислотная последовательность megaTAL, которая связывает и расщепляет сайт-мишень в гене PD-1 человека.
SEQ ID NO: 16 - аминокислотная последовательность megaTAL, которая связывает и расщепляет сайт-мишень в гене PD-1 человека.
SEQ ID NO: 17 - аминокислотная последовательность megaTAL, которая связывает и расщепляет сайт-мишень в гене PD-1 человека.
SEQ ID NO: 18 - аминокислотная последовательность megaTAL, которая связывает и расщепляет сайт-мишень в гене PD-1 человека.
SEQ ID NO: 19 - аминокислотная последовательность megaTAL, которая связывает и расщепляет сайт-мишень в гене PD-1 человека.
SEQ ID NO: 20 - аминокислотная последовательность megaTAL, которая связывает и расщепляет сайт-мишень в гене PD-1 человека.
SEQ ID NO: 21 - аминокислотная последовательность megaTAL, которая связывает и расщепляет сайт-мишень в гене PD-1 человека.
SEQ ID NO: 22 - аминокислотная последовательность megaTAL, которая связывает и расщепляет сайт-мишень в гене PD-1 человека.
SEQ ID NO: 23 - аминокислотная последовательность megaTAL, которая связывает и расщепляет сайт-мишень в гене PD-1 человека.
SEQ ID NO: 24 - аминокислотная последовательность, кодирующая мышиный Trex2.
SEQ ID NO: 25 - сайт-мишень варианта LHE I-OnuI в экзоне 5 гена PD-1 человека.
SEQ ID NO: 26 - сайт-мишень ДНК-связывающего домена TALE в экзоне 5 гена PD-1 человека.
SEQ ID NO: 27 - сайт-мишень megaTAL в экзоне 5 гена PD-1 человека.
SEQ ID NO: 28 - сайт-мишень варианта I-OnuI LHE N-концевого домена в экзоне 5 гена PD-1 человека.
SEQ ID NO: 29 - сайт-мишень варианта I-OnuI LHE C-концевого домена в экзоне 5 гена PD-1 человека.
SEQ ID NO: 30 - сайт-мишень варианта I-OnuI LHE в экзоне 1 гена PD-1 человека.
SEQ ID NO: 31 - сайт-мишень ДНК-связывающего домена TALE в экзоне 1 гена PD-1 человека.
SEQ ID NO: 32 - сайт-мишень megaTAL в экзоне 1 гена PD-1 человека.
SEQ ID NO: 33 - сайт-мишень варианта I-OnuI LHE N-концевого домена в экзоне 1 гена PD-1 человека.
SEQ ID NO: 34 - сайт-мишень варианта I-OnuI LHE C-концевого домена в экзоне 1 гена PD-1 человека.
SEQ ID NO: 35 - сайт-мишень варианта I-OnuI LHE в экзоне 2 гена PD-1 человека.
SEQ ID NO: 36 - сайт-мишень ДНК-связывающего домена TALE в экзоне 2 гена PD-1 человека.
SEQ ID NO: 37 - сайт-мишень megaTAL в экзоне 2 гена PD-1 человека.
SEQ ID NO: 38 - сайт-мишень варианта I-OnuI LHE N-концевого домена в экзоне 2 гена PD-1 человека.
SEQ ID NO: 39 - сайт-мишень варианта I-OnuI LHE C-концевого домена в экзоне 2 гена PD-1 человека.
SEQ ID NO: 40 - мРНК, кодирующая megaTAL PD-1.
SEQ ID NO: 41 - мРНК, кодирующая megaTAL PD-1.
SEQ ID NO: 42 - мРНК, кодирующая megaTAL PD-1.
SEQ ID NO: 43 - мРНК, кодирующая мышиный белок Trex2.
SEQ ID NO: 44 - полинуклеотидная последовательность, кодирующая нацеленный на rAAV вектор с гомологичными плечами PD-1, и кассета экспрессии pMND-BFP-SV40polyA.
SEQ ID NO: 45 - полинуклеотид, кодирующий 5'-гомологичное плечо SEQ ID NO: 44.
SEQ ID NO: 46 - полинуклеотид, кодирующий 3'-гомологичное плечо SEQ ID NO: 44.
SEQ ID NO: 47 - полинуклеотидная последовательность, кодирующая нацеленный на rAAV вектор с гомологичными плечами PD-1, и кассета экспрессии pMND-BFP-SV40polyA. Плечо 3' содержит одиночный нуклеотидный полиморфизм (SNP) относительно геномной последовательности дикого типа.
SEQ ID NO: 48 - полинуклеотидная последовательность, кодирующая нацеленный на rAAV вектор с гомологичными плечами PD-1, и кассета экспрессии pMND-BFP-SV40polyA. Плечо 5' содержит одиночный нуклеотидный полиморфизм (SNP) относительно геномной последовательности дикого типа.
SEQ ID NO: 49 - полинуклеотид, кодирующий 5'- гомологичное плечо SEQ ID NO: 48.
SEQ ID NO: 50 - полинуклеотид, кодирующий 3'- гомологичное плечо SEQ ID NO: 47.
SEQ ID NO: 51 - полинуклеотидная последовательность, кодирующая вектор, нацеленный на rAAV, с кассетой экспрессии pMND-PD-1.CD28. переключающий рецептор SV40polyA.
SEQ ID NO: 52 - полинуклеотидная последовательность, кодирующая нацеленный на rAAV вектор с гомологичными плечами PD-1, и кассету экспрессии pMND-BFP.SV40polyA с 5'-гомологичным плечом ~1,3 т.п.н. в обратном направлении от мотива ITSM в экзоне 5 PD-1.
SEQ ID NO: 53 - полинуклеотидная последовательность, кодирующая нацеленный на rAAV вектор с гомологичными плечами PD-1, и кассету экспрессии pMND-GFP-SV40polyA.
SEQ ID NO: 54 - полинуклеотид, кодирующий 5'-гомологичное плечо SEQ ID NO: 53.
SEQ ID NO: 55 - полинуклеотид, кодирующий 3'-гомологичное плечо SEQ ID NO: 53.
SEQ ID NO: 56 - полинуклеотидная последовательность, кодирующая нацеленный на rAAV вектор с гомологичными плечами PD-1, и кассету экспрессии pMND-анти-CD19 CAR-SV40polyA.
SEQ ID NO: 57 - полинуклеотидная последовательность, кодирующая нацеленный на rAAV вектор с гомологичными плечами PD-1, и кассету экспрессии pMND-анти-BCMA CAR-SV40polyA.
SEQ ID NO: 58 - полинуклеотидная последовательность, кодирующая нацеленный на rAAV вектор с гомологичными плечами PD-1, и кДНК, кодирующая mCherry.
SEQ ID NO: 60 - аминокислотная последовательность варианта LHE I-OnuI, перепрограммированного для связывания и расщепления сайта-мишени в гене PD-1 человека
SEQ ID NO: 61 - аминокислотная последовательность варианта LHE I-OnuI, перепрограммированного для связывания и расщепления сайта-мишени в гене PD-1 человека
SEQ ID NO: 62 - аминокислотная последовательность варианта LHE I-OnuI, перепрограммированного для связывания и расщепления сайта-мишени в гене PD-1 человека
SEQ ID NO: 63 - аминокислотная последовательность варианта LHE I-OnuI, перепрограммированного для связывания и расщепления сайта-мишени в гене PD-1 человека
SEQ ID NO: 64 - аминокислотная последовательность megaTAL, которая связывает и расщепляет сайт-мишень в гене PD-1 человека
SEQ ID NO: 65 - мРНК, кодирующая megaTAL PD-1
SEQ ID NO: 66 - мРНК, кодирующая megaTAL PD-1
SEQ ID NO: 67 - мРНК, кодирующая megaTAL PD-1
SEQ ID NO: 68 - мРНК, кодирующая megaTAL PD-1
SEQ ID NO: 69-79 - аминокислотные последовательности различных линкеров.
SEQ ID NO: 80-104 - аминокислотные последовательности сайтов расщепления протеазой и сайтов расщепления саморасщепляемого полипептида.
В приведенных выше последовательностях символ X, где он присутствует, обозначает любую аминокислоту или отсутствие аминокислоты.
Подробное описание
A. ОБЗОР
Настоящее изобретение в целом относится, в частности, к улучшенным композициям для редактирования генома и способам их применения. Без привязки к какой-либо конкретной теории, композиции для редактирования генома, рассматриваемые в различных вариантах реализации, можно использовать для предотвращения или лечения рака, инфекционного заболевания, аутоиммунного заболевания, воспалительного заболевания и иммунодефицита или состояния, ассоциированного с ними, или для облегчения по меньшей мере одного их симптома. Одним из ограничений или проблем, которые являются слабым местом существующей адоптивной клеточной терапии, является гипореактивность иммунных эффекторных клеток вследствие истощения, опосредованного микроокружением опухоли. Истощенные Т-клетки имеют уникальную молекулярную сигнатуру, которая заметно отличается от наивных, эффекторных Т-клеток или Т-клеток памяти. Истощенные Т-клетки определяются как Т-клетки со снижением экспрессии цитокинов и эффекторной функции. PD-1 является маркером истощения Т-клеток; повышенная экспрессия PD-1 ассоциирована со снижением пролиферации Т-клеток и снижением выработки IL-2, TNF и IFN-γ.
В частных вариантах реализации рассматриваемые в настоящей заявке иммунные эффекторные клетки с отредактированным геномом становятся более устойчивыми к истощению за счет устранения, уменьшения или подавления экспрессии и/или передачи сигналов PD-1.
Композиции и способы редактирования генома, рассматриваемые в различных вариантах реализации, содержат варианты нуклеаз, предназначенные для связывания и расщепления сайта-мишени в гене запрограммированной клеточной гибели 1 (PD-1). Варианты нуклеазы, рассматриваемые в частных вариантах реализации могут быть применены для введения двухцепочечного разрыва в целевую полинуклеотидную последовательность, которая может быть подвернута репарации негомологичным соединением концов (NHEJ) в отсутствие полинуклеотидной матрицы, например, донорной матрицы репарации, или гомологичной направленной репарацией (HDR), т.е. гомологичной рекомбинацией, в присутствии донорной матрицы репарации. Варианты нуклеазы, рассматриваемые в некоторых вариантах реализации, также могут быть сконструированы в виде никаз, генерирующих одноцепочечные разрывы ДНК, которые могут быть репарированы с использованием механизма клеточного эксцизионной репарации оснований (BER) или гомологичной рекомбинации в присутствии донорной матрицы репарации. NHEJ является допускающим ошибки процессом, который часто приводит к образованию небольших инсерций и делеций, которые нарушают функцию генов. Гомологичная рекомбинация требует гомологичной ДНК в качестве матрицы для репарации и может быть использована для создания неограниченного разнообразия модификаций, определяемых введением донорной ДНК, содержащей желаемую последовательность на сайте-мишени, фланкированную с обеих сторон последовательностями, несущими гомологию с областями, фланкирующими сайт-мишень.
В одном предпочтительном варианте реализации рассматриваемые в настоящей заявке композиции для редактирования генома содержат вариант хоминг-эндонуклеазы или megaTAL, который нацелен на ген PD-1 человека.
В одном предпочтительном варианте реализации рассматриваемые в настоящей заявке композиции для редактирования генома содержат вариант хоминг-эндонуклеазы или megaTAL и фермент, изменяющий структуру концов, например Trex2.
В различных вариантах реализации рассматриваются клетки с отредактированным геномом. Клетки с отредактированным геномом содержат отредактированный ген PD-1, причем стратегия редактирования предназначена для уменьшения или устранения экспрессии PD-1 и/или кооптирования PD-1, чтобы он действовал в качестве доминантного отрицательного, путем экспрессии внеклеточного лиганд-связывающего домена PD-1, при нарушении его способности трансдуцировать иммуносупрессивные внутриклеточные сигналы.
В различных вариантах реализации создают разрыв ДНК в сайте-мишени гена PD-1 в Т-клетке, например, в иммунной эффекторной клетке, и NHEJ концов расщепленной геномной последовательности может привести к появлению клетки практически без экспрессии PD-1 и, предпочтительно, Т-клетки, в которой отсутствует или практически отсутствует функциональная экспрессия и/или передача сигналов PD-1, например, отсутствует способность увеличивать истощение Т-клеток. Без привязки к какой-либо конкретной теории, Т-клетки, у которых отсутствует функциональная экспрессия PD-1, более устойчивы к иммуносупрессии и истощению Т-клеток и, следовательно, более устойчивы и терапевтически эффективны.
В различных других вариантах реализации предусмотрена донорная матрица для репарации расщепленной геномной последовательности PD-1. Ген PD-1 репарируют с помощью последовательности указанной матрицы путем гомологичной рекомбинации в сайте разрыва ДНК. В частных вариантах реализации матрица репарации содержит полинуклеотидную последовательность, которая нарушает и предпочтительно существенно снижает или устраняет функциональную экспрессию PD-1.
В частных вариантах реализации ген PD-1 репарируют с помощью матрицы, кодирующей экзодомен PD-1 с повышенной аффинностью к его лигандам.
В частных вариантах реализации ген PD-1 репарируют полинуклеотидом, кодирующим энхансер иммунопотенции, демпфер иммуносупрессивного сигнала или сконструированный рецептор антигена.
В частных вариантах реализации ген PD-1 репарируют полинуклеотидом, кодирующим энхансер иммунопотенции, демпфер иммуносупрессивного сигнала или сконструированный рецептор антигена, и вводят в ген PD-1 для кооптирования эндогенного промотора PD-1 с целью транскрипционного контроля экспрессии энхансера иммунопотенции, демпфера иммуносупрессивного сигнала или сконструированного рецептора антигена.
В предпочтительных вариантах реализации композиции генома и способы для редактирования, рассматриваемые в настоящей заявке, используют для редактирования гена PD-1 человека.
Соответственно, способы и композиции, рассматриваемые в настоящей заявке, представляют собой принципиальное улучшение по сравнению с существующими адаптивными клеточными методами лечения.
В практике частных вариантов реализации будут использованы, если не указано иное, традиционные методы химии, биохимии, органической химии, молекулярной биологии, микробиологии, рекомбинантной ДНК, генетики, иммунологии и клеточной биологии, которые известны специалистам в данной области, многие из которых описаны ниже с целью иллюстрации. Такие методики подробно описаны в литературе. Смотрите, например, Sambrook, et al., Molecular Cloning: A Laboratory Manual (3rd Edition, 2001); Sambrook, et al., Molecular Cloning: A Laboratory Manual (2nd Edition, 1989); Maniatis et al., Molecular Cloning: A Laboratory Manual (1982); Ausubel et al., Current Protocols in Molecular Biology (John Wiley and Sons, updated July 2008); Short Protocols in Molecular Biology: A Compendium of Methods from Current Protocols in Molecular Biology, Greene Pub. Associates and Wiley-Interscience; Glover, DNA Cloning: A Practical Approach, vol. I & II (IRL Press, Oxford, 1985); Anand, Techniques for the Analysis of Complex Genomes, (Academic Press, New York, 1992); Transcription and Translation (B. Hames & S. Higgins, Eds., 1984); Perbal, A Practical Guide to Molecular Cloning (1984); Harlow and Lane, Antibodies, (Cold Spring Harbor Laboratory Press, Cold Spring Harbor, N.Y., 1998) Current Protocols in Immunology Q. E. Coligan, A. M. Kruisbeek, D. H. Margulies, E. M. Shevach and W. Strober, eds., 1991); Annual Review of Immunology; а также работы в журналах, таких как Advances in Immunology.
B. ОПРЕДЕЛЕНИЯ
Если не указано иное, все технические и научные термины, используемые в настоящей заявке, имеют то же значение, которое обычно понимают специалисты в области техники, к которой относится изобретение. Хотя при практическом применении или испытании частных вариантов реализации могут использоваться любые способы и материалы, подобные или эквивалентные тем, которые описаны в настоящей заявке, в настоящей заявке описаны предпочтительные варианты реализации композиций, способов и материалов. Для целей настоящего изобретения ниже определены следующие термины.
Определенные и неопределенные артикли используются в настоящей заявке для обозначения одного или более чем одного (то есть по меньшей мере одного или одного или нескольких) грамматического объекта. Например, «элемент» обозначает один элемент или один или более элементов.
Использование альтернативных вариантов (например, «или») следует понимать как означающее либо один, либо оба, либо любую комбинацию данных альтернативных вариантов.
Термин «и/или» следует понимать как означающий либо один, либо оба альтернативных варианта.
Используемый в настоящей заявке термин «приблизительно» или «около» относится к количеству, уровню, значению, числу, частоте, проценту, размеру, габариту, количеству, весу или длине, которые варьируются на 15%, 10%, 9. %, 8%, 7%, 6%, 5%, 4%, 3%, 2% или 1% относительно контрольного количества, уровня, значения, числа, частоты, процента, размера, габарита, количества, веса или длины. В одном варианте реализации термин «приблизительно» или «около» относится к диапазону количества, уровня, значения, числа, частоты, процента, габарита, размера, количества, веса или длины ±15%, ±10%, ±9%, ±8%, ±7%, ±6%, ±5%, ±4%, ±3%, ±2% или ±1% относительно эталонной величины, уровня, значения, числа, частоты, процента, габарита, размера, количества, веса или длины.
В одном варианте реализации диапазон, например от 1 до 5, от приблизительно 1 до 5 или от приблизительно 1 до приблизительно 5, относится к каждому числовому значению, охватываемому этим диапазоном. Например, в одном неограничивающем и исключительно иллюстративном варианте реализации диапазон от 1 до 5 эквивалентен выражению 1, 2, 3, 4, 5; или 1.0, 1.5, 2.0, 2.5, 3.0, 3.5, 4.0, 4.5, или 5.0; или 1.0, 1.1, 1.2, 1.3, 1.4, 1.5, 1.6, 1.7, 1.8, 1.9, 2.0, 2.1, 2.2, 2.3, 2.4, 2.5, 2.6, 2.7, 2.8, 2.9, 3.0, 3.1, 3.2, 3.3, 3.4, 3.5, 3.6, 3.7, 3.8, 3.9, 4.0, 4.1, 4.2, 4.3, 4.4, 4.5, 4.6, 4.7, 4.8, 4.9, или 5.0.
Используемый в настоящей заявке термин «по существу» относится к количеству, уровню, значению, числу, частоте, проценту, размеру, габариту, количеству, весу или длине, которые составляют 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или выше по сравнению с эталонной величиной, уровнем, значением, числом, частотой, процентом, габаритом, размером, количеством, весом или длиной. В одном варианте реализации «по существу, то же самое» относится к количеству, уровню, значению, числу, частоте, проценту, габариту, размеру, количеству, весу или длине, которые производят эффект, например, физиологический эффект, который приблизительно такой же, как эталонная величина, уровень, значение, число, частота, процент, габарит, размер, количество, вес или длина.
Во всем описании, если контекст не требует иного, слова «содержать», «содержит» и «содержащий» будут пониматься как подразумевающие включение указанного этапа или элемента или группы этапов или элементов, но не исключение какого-либо другого этапа или элемента или группы этапов или элементов. Термин «состоящий из» означает включающий все и ограниченный всем, что следует за фразой «состоящий из». Таким образом, фраза «состоящий из» означает, что перечисленные элементы являются обязательными или необходимыми и что никакие другие элементы присутствовать не могут. Термин «состоящий в основном из» означает включающий любые элементы, перечисленных после фразы, и ограниченный другими элементами, которые не мешают или способствуют активности или действию, указанным в описании для перечисленных элементов. Таким образом, фраза «состоящий в основном из» указывает на то, что перечисленные элементы являются обязательными или необходимыми, но другие элементы, которые существенным образом влияют на активность или действие перечисленных элементов, отсутствуют.
Ссылка по всему описанию на «один вариант реализации», «вариант реализации», «частный вариант реализации», «связанный вариант реализации», «некоторый вариант реализации», «дополнительный вариант реализации» или «другой вариант реализации» или их комбинации означает, что конкретный признак, структура или характеристика, описанные в связи с этим вариантом реализации, включены по меньшей мере в один вариант реализации. Таким образом, появления вышеупомянутых фраз в различных местах в настоящем описании не обязательно все относятся к одному и тому же варианту реализации. Кроме того, конкретные признаки, структуры или характеристики могут быть объединены любым подходящим способом в одном или нескольких вариантах реализации. Также понятно, что положительное включение признака в одном варианте реализации служит основанием для исключения указанного признака в частном варианте реализации.
Термин «ex vivo» обычно относится к действиям, которые происходят вне организма, таким как эксперименты или измерения, проводимые в или на живых тканях в искусственной среде вне организма, предпочтительно с минимальным изменением естественных условий. В частных вариантах реализации процедуры «ex vivo» включают живые клетки или ткани, взятые из организма и культивируемые или изменяемые в лабораторном аппарате, обычно в стерильных условиях и обычно в течение нескольких часов или приблизительно до 24 часов, но в том числе до 48 или 72 часа, в зависимости от обстоятельств. В некоторых вариантах реализации такие ткани или клетки можно собирать и замораживать, а затем размораживать для обработки ex vivo. Эксперименты или процедуры в области культивирования тканей, которые длятся дольше, чем несколько дней, с использованием живых клеток или тканей, как правило, считаются «in vitro», хотя в некоторых вариантах реализации указанный термин может использоваться взаимозаменяемо с «ex vivo».
Термин «in vivo» в целом относится к деятельности, которая происходит внутри организма. В одном варианте реализации клеточные геномы являются сконструированными, отредактированными или модифицированными in vivo.
Термины «усиливать» или «способствовать» или «увеличивать», «расширять» или «потенцировать» обычно относятся к способности варианта нуклеазы, композиции для редактирования генома или клетки с отредактированным геномом, рассматриваемых в настоящей заявке, производить или вызывать больший ответ (то есть, физиологический ответ) по сравнению с ответом, вызванным пустым носителем или контролем. Измеримый ответ может включать в себя увеличение каталитической активности, аффинности связывания, устойчивости, цитолитической активности и/или повышение провоспалительных цитокинов, среди прочих эффектов, очевидных из понимания в данной области техники и описания в настоящей заявке. «Увеличенное» или «расширенное» количество обычно представляет собой «статистически значимое» количество и может включать увеличение в 1,1, 1,2, 1,5, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20, 30 или более раз (например, 500, 1000 раз) (включая все целые числа и десятичные знаки между ними и выше 1, например, 1,5, 1,6, 1,7, 1,8 и т.д.) по сравнению с ответом, производимым пустым носителем или контролем.
Термины «уменьшать» или «снижать» или «сокращать» или «ослаблять» или «ингибировать», или «подавлять» или «амортизировать» обычно обозначают способность варианта нуклеазы, композиции для редактирования генома или клетки с отредактированным геномом, рассматриваемых в настоящей заявке, вызывает, производить или вызывать меньший ответ (то есть физиологический ответ) по сравнению с ответом, вызванным пустым носителем, либо контролем. Измеримый ответ может включать в себя снижение нецелевой связывающей аффинности, нецелевой специфичности расщепления, истощения Т-клеток и тому подобного. «Уменьшенное» или «сниженное» количество обычно представляет собой «статистически значимое» количество и может включать уменьшение в 1,1, 1,2, 1,5, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15., 20, 30 или более раз (например, 500, 1000 раз) (включая все целые и десятичные знаки между ними и выше 1, например, 1,5, 1,6, 1,7, 1,8 и т.д.) по сравнению с ответом (эталонный ответ), производимый пустым носителем или контролем.
Термины «поддерживать», «сохранять», «поддержание», «без изменений», «без существенных изменений» или «без существенного уменьшения» обычно обозначают способность варианта нуклеазы, композиции для редактирования генома или клетки с отредактированным геномом, рассматриваемых в настоящей заявке, производит, производить или вызывать по существу сходный или сопоставимый физиологический ответ (т.е. эффекты в направлении по ходу транскрипции) по сравнению с ответом, вызванным либо пустым носителем, либо контролем. Сравнимый ответ - это ответ, который существенно не отличается или не отличается измеримо от контрольного ответа.
Термины «специфическая аффинность связывания» или «специфически связывает», или «специфически связанный», или «специфическое связывание», или «специфически нацеленный», используемые в настоящей заявке, описывают связывание одной молекулы с другой, например, ДНК-связывающий домен полипептида, связывающегося с ДНК, с большей аффинностью связывания, чем фоновое связывание. Домен связывания «специфически связывается» с сайтом-мишенью, если он связывается с сайтом-мишенью или связывается с ним с аффинностью или Ka (т.е. равновесной константой ассоциации конкретного связывающего взаимодействия с единицами 1/M) величиной, например, больше или равной приблизительно 105 М-1. В некоторых вариантах реализации связывающий домен связывается с сайтом-мишенью с Ka, больше или равной приблизительно 106 M-1, 107 M-1, 108 M-1, 109 M-1, 1010 M-1, 1011 M-1, 1012 M-1 или 1013 M-1. Связывающие домены «с высокой аффинностью» обозначают связывающие домены с Ka величиной по меньшей мере 107 M-1, по меньшей мере 108 M-1, по меньшей мере 109 M-1, по меньшей мере 1010 M-1, по меньшей мере 1011 M-1, по меньшей мере 1012 M-1, по меньшей мере 1013 M-1, или больше.
В альтернативном варианте аффинность может быть определена как константа диссоциации равновесия (Kd) конкретного связывающего взаимодействия с единицами M (например, от 10-5 M до 10-13 M, или менее). Аффинность вариантов нуклеазы, включающих один или более ДНК-связывающих доменов для сайтов-мишеней ДНК, рассматриваемых в частных вариантах реализации может быть легко определена с применением общепринятых методов, например, представления на поверхности дрожжевых клеток, или анализа ассоциации связывания или анализа замещения с использованием меченых лигандов.
В одном варианте реализации аффинность специфического связывания приблизительно в 2 раза больше, чем фоновое связывание, приблизительно в 5 раз больше, чем фоновое связывание, приблизительно в 10 раз больше, чем фоновое связывание, приблизительно в 20 раз больше, чем фоновое связывание, приблизительно в 50 раз больше, чем фоновое связывание, приблизительно в 100 раз больше, чем фоновое связывание, или приблизительно в 1000 раз больше, чем фоновое связывание, или еще больше.
Термины «избирательно связывает» или «избирательно связанный, или «избирательное связывание», или «избирательно нацеленный» описывают преимущественное связывание одной молекулы с молекулой-мишенью (целевое связывание) в присутствии множества внецелевых молекул. В частных вариантах реализации HE или megaTAL избирательно связывают сайт целевого связывания ДНК приблизительно в 5, 10, 15, 20, 25, 50, 100 или 1000 раз чаще, чем HE или megaTAL связывают сайт связывания внецелевой ДНК.
«Целевой» относится к последовательности сайта-мишени.
«Вне-целевой» относится к последовательности, аналогичной, но не идентичной последовательности сайта-мишени.
«Сайт-мишень» или «целевая последовательность» представляет собой последовательность хромосомной или внехромосомной нуклеиновой кислоты, которая определяет часть нуклеиновой кислоты, которую связывающая молекула будет связывать и/или расщеплять, при условии наличия достаточных условий для связывания и/или расщепления. При упоминании полинуклеотидной последовательности или SEQ ID NO со ссылкой только на одну цепь сайта-мишени или последовательности-мишени, следует понимать, что эти сайт-мишень или последовательность-мишень, связанные и/или расщепленные вариантом нуклеазы, являются двухцепочечными и содержат эталонную последовательность и ее комплемент. В предпочтительном варианте сайт-мишень представляет собой последовательность в гене PD-1 человека.
«Рекомбинация» относится к процессу обмена генетической информацией между двумя полинуклеотидами, включая, помимо прочего, захват доноров негомологичным соединением концов (NHEJ) и гомологичную рекомбинацию. Для целей настоящего изобретения термин «гомологичная рекомбинация (HR)» относится к специализированной форме такого обмена, который имеет место, например, во время репарации двухцепочечных разрывов в клетках посредством механизмов гомологичной репарации (HDR). Указанный процесс требует гомологии нуклеотидной последовательности, использует «донорную» молекулу в качестве матрицы для восстановления «целевой» молекулы (то есть той, которая испытала двухцепочечный разрыв) и в разных вариантах известен как «генная конверсия без кроссовера» или «генная конверсия короткого тракта», потому что он приводит к передаче генетической информации от донора к мишени. Без привязки к какой-либо конкретной теории, такой перенос может включать коррекцию несоответствия гетеродуплексной ДНК, которая образуется между поврежденной мишенью и донором, и/или «синтез-зависимый отжиг цепи», при котором используют донор для ресинтеза генетической информации. которая станет частью цели и/или связанных процессов. Такая специализированная HR часто приводит к изменению последовательности молекулы-мишени, так что часть или вся последовательность донорного полинуклеотида включается в полинуклеотид-мишень.
Термин «NHEJ» или «негомологичное соединение концов» относится к разрешению двухцепочечного разрыва в отсутствие донорной матрицы репарации или гомологичной последовательности. NHEJ может привести к инсерциям и делециям на месте разрыва. NHEJ опосредуется несколькими суб-путями, каждый из которых имеет явные мутационные последствия. Классический путь NHEJ (cNHEJ) требует комплекса KU/DNA-PKcs/Lig4/XRCC4, снова лигирует концы вместе с минимальным процессингом и часто приводит к точной репарации разрыва. Альтернативные пути NHEJ (altNHEJ) также активны в разрешении разрывов дцДНК, но эти пути являются значительно более мутагенными и часто приводят к неточной репарации разрыва, отмеченной инсерциями и делециями. Без привязки к какой-либо конкретной теории, предполагается, что модификация разрывов дцДНК ферментами, изменяющими структуру концов, такими как, например, экзонуклеазы, например, Trex2, может увеличить вероятность неточной репарации.
«Расщепление» относится к разрушению ковалентного остова молекулы ДНК. Расщепление может быть инициировано различными способами, включая, среди прочего, ферментативный или химический гидролиз фосфодиэфирной связи. Возможно как одноцепочечное расщепление и двухцепочечное расщепление. Двухцепочечное расщепление может происходить в результате двух отдельных событий одноцепочечного расщепления. Расщепление ДНК может привести к образованию либо тупых концов, либо ступенчатых концов. В некоторых вариантах реализации полипептиды и варианты нуклеаз, например варианты хоминг-эндонуклеазы, megaTAL и др., рассматриваемые в настоящей заявке, используются для направленного двухцепочечного расщепления ДНК. Сайты распознавания эндонуклеазного расщепления могут находиться на любой цепи ДНК.
«Экзогенная» молекула - это молекула, которая обычно не присутствует в клетке, но которая вводится в клетку одним или несколькими генетическими, биохимическими или другими методами. Типичные экзогенные молекулы включают, среди прочего, небольшие органические молекулы, белок, нуклеиновую кислоту, углевод, липид, гликопротеин, липопротеин, полисахарид, любое модифицированное производное вышеуказанных молекул или любой комплекс, включающий одну или более вышеуказанных молекул. Способы введения экзогенных молекул в клетки известны специалистам в данной области и включают, среди прочего, липид-опосредованный перенос (т.е. липосомы, включая нейтральные и катионные липиды), электропорацию, прямую инъекцию, слияние клеток, бомбардировка частицами, наночастицы биополимера, соосаждение фосфата кальция, DEAE-декстран-опосредованный перенос и опосредованный вирусным вектором переносом.
«Эндогенная» молекула - это молекула, которая обычно присутствует в конкретной клетке на определенной стадии развития в определенных условиях окружающей среды. Дополнительно эндогенные молекулы могут включать белки.
«Ген» относится к области ДНК, кодирующей продукт гена, а также ко всем областям ДНК, которые регулируют выработку продукта гена, независимо от того, соседствуют ли такие регуляторные последовательности с кодирующими и/или транскрибируемыми последовательностями. Ген содержит, среди прочего, промоторные последовательности, энхансеры, сайленсеры, инсуляторы, граничные элементы, терминаторы, последовательности полиаденилирования, посттранскрипционные ответные элементы, трансляционные регуляторные последовательности, такие как сайты связывания рибосом и внутренние сайты входа рибосом, источники репликации, сайты прикрепления матрицы и области локусов контроля.
«Экспрессия гена» относится к преобразованию информации, содержащейся в гене, в продукт гена. Продукт гена может быть прямым продуктом транскрипции гена (например, мРНК, тРНК, рРНК, антисмысловая РНК, рибозима, структурная РНК или РНК любого другого типа) или белком, полученным путем трансляции мРНК. Продукты гена также включают РНК, которые модифицированы такими процессами, как кэппирование, полиаденилирование, метилирование и редактирование, и белками, модифицированными, например, метилированием, ацетилированием, фосфорилированием, убиквитинированием, АДФ-рибозилированием, миристилированием и гликозилированием.
Используемый в настоящей заявке термин «сконструированный» или «генетически модифицированный» относится к хромосомному или внехромосомному добавлению дополнительного генетического материала в форме ДНК или РНК к общему генетическому материалу в клетке. Генетические модификации могут быть нацеленными или не нацеленными на конкретный сайт в геноме клетки. В одном варианте реализации генетическая модификация является сайт-специфичной. В другом варианте реализации генетическая модификация не является сайт-специфичной.
Используемый в настоящей заявке термин «редактирование генома» относится к замене, делеции и/или введению генетического материала в сайт-мишень в геноме клетки, которые восстанавливают, исправляют, нарушают и/или модифицируют экспрессию и/или функцию гена или продукта гена. Редактирование генома, предлагаемое в частных вариантах реализации включает в себя введение одного или нескольких вариантов нуклеазы в клетку для генерирования повреждений ДНК на или вблизи от сайта-мишени в геноме клетки, необязательно в присутствии донорной матрицы репарации.
Используемый в настоящей заявке термин «генная терапия» относится к введению дополнительного генетического материала в общий генетический материал в клетке, которое восстанавливает, корректирует или модифицирует экспрессию гена или продукта гена, либо с целью экспрессии терапевтического полипептида. В частных вариантах реализации введение генного материала в геном клетки путем редактирования генома, которое восстанавливает, корректирует, нарушает или изменяет экспрессию гена или продукта гена, либо с целью экспрессии терапевтического полипептида считается генной терапией.
«Иммунное расстройство» относится к заболеванию, которое вызывает реакцию иммунной системы. В частных вариантах реализации термин «иммунное расстройство» относится к раку, болезни «трансплантат против хозяина», аутоиммунному заболеванию или иммунодефициту. В одном варианте реализации иммунные нарушения охватывают инфекционное заболевание.
Используемый в настоящей заявке термин «рак» обычно относится к классу заболеваний или состояний, при которых аномальные клетки делятся без контроля и могут проникать в близлежащие ткани.
Используемый в настоящей заявке термин «злокачественный» относится к раку, при котором группа опухолевых клеток демонстрирует одно или более из: неконтролируемого роста (т.е. деления за рамки нормальных пределов), инвазии (то есть вторжения в соседние ткани и их разрушения) и метастазирования (т.е. распространение в другие участки тела через лимфу или кровь).
Используемый в настоящей заявке термин «метастазирование» относится к распространению рака из одной части тела в другую. Опухоль, образованная распространяющимися клетками, называется «метастатическая опухоль» или «метастаз». Метастатическая опухоль содержит клетки, подобные клеткам в исходной (первичной) опухоли.
Используемый в настоящей заявке термин «доброкачественный» или «незлокачественный» относится к опухолям, которые могут увеличиваться в размерах, но не распространяться на другие части тела. Доброкачественные опухоли самоограничены и обычно не проникают в другие участки и не метастазируют.
«Раковая клетка» или «опухолевая клетка» относится к отдельной клетке ракового роста или ткани. «Опухоль», как правило, относится к отеку или поражению, образованному аномальным ростом клеток, которые могут быть доброкачественными, предзлокачественными или злокачественными. Большинство видов рака образуют опухоли, но некоторые, например, лейкемия, не обязательно образуют опухоли. Для тех видов рака, которые образуют опухоли, термины рак (раковая клетка) и опухоль (опухолевая клетка) используются взаимозаменяемо. Количество опухоли у индивидуума представляет собой «опухолевую нагрузку», которое можно измерить как количество, объем или вес опухоли.
Термин «болезнь трансплантат против хозяина» или «БТПХ» относится к осложнениям, которые могут возникнуть после трансплантации клетки, ткани или солидного органа. БТПХ может возникнуть после трансплантации стволовых клеток или костного мозга, при которой трансплантированные донорские клетки атакуют тело реципиента трансплантата. Острая БТПХ у людей возникает в течение приблизительно 60 дней после трансплантации и приводит к поражению кожи, печени и кишечника под действием цитолитических лимфоцитов. Хроническая БТПХ возникает позже и представляет собой системное аутоиммунное заболевание, которое поражает главным образом кожу, что приводит к поликлональной активации В-клеток и гиперпродукции Ig и аутоантител. Болезнь «трансплантат против хозяина» трансплантата солидного органа (SOT-БТПХ) встречается в двух формах. Более распространенным типом является опосредованное антителами, при котором антитела донора с группой крови O атакуют эритроциты реципиента у реципиентов с группой крови A, B или AB, что приводит к легким преходящим гемолитическим анемиям. Вторая форма SOT-БТПХ представляет собой клеточный тип, ассоциированный с высокой смертностью, когда донорские Т-клетки вызывают иммунологическую атаку против иммунологически несовместимой ткани хозяина, чаще всего на коже, печени, желудочно-кишечном тракте и костном мозге, что приводит к осложнениям в этих органах.
Термин «трансплантат против лейкоза» или «GVL» относится к иммунному ответу на клетки лейкоза человека посредством иммунных клеток, присутствующих в трансплантированной ткани донора, такой как костный мозг или периферическая кровь.
«Аутоиммунное заболевание» относится к заболеванию, при котором организм производит иммуногенный ответ (т.е. ответ иммунной системы) на некоторую составляющую своей собственной ткани. Другими словами, иммунная система теряет способность распознавать какую-то ткань или систему в организме как «собственные» и нацеливается на нее и атакует ее, как если бы она была чужеродной. Иллюстративные примеры аутоиммунных заболеваний включают, среди прочего: артрит, воспалительное заболевание кишечника, тиреоидит Хашимото, болезнь Грейвса, волчанку, рассеянный склероз, ревматический артрит, гемолитическую анемию, антииммунный тиреоидит, системную красную волчанку, целиакию, болезнь Крона, колит, диабет, склеродермию, псориаз и тому подобное.
«Иммунодефицит» означает состояние пациента, иммунная система которого была нарушена в результате заболевания или применения химикатов. Это состояние делает систему дефицитной по количеству и типу клеток крови, необходимых для защиты от инородных субстанций. Состояния или заболевания иммунодефицита известны в данной области и включают, например, СПИД (синдром приобретенного иммунодефицита), SCID (заболевание тяжелого комбинированного иммунодефицита), селективный дефицит IgA, общий вариабельный иммунодефицит, Х-сцепленную агаммаглобулинемию, хроническую гранулематозную болезнь, гипер-IgM синдром Вискотта-Олдрича (WAS) и диабет.
«Инфекционное заболевание» относится к заболеванию, которое может передаваться от человека к человеку или от организма к организму и вызывается микробным или вирусным агентом (например, вирусом простуды). Инфекционные заболевания известны в данной области и включают, например, гепатит, заболевания, передаваемые половым путем (например, хламидиоз, гонорея), туберкулез, ВИЧ/СПИД, дифтерию, гепатит B, гепатит C, холеру и грипп.
Используемые в настоящей заявке термины «индивидуум» и «субъект» часто используются взаимозаменяемо и относятся к любому животному, у которого проявляется симптом иммунного нарушения, которое можно лечить с помощью вариантов нуклеазы, композиций для редактирования генома, векторов для генной терапии, векторов для редактирования генома, клеток с отредактированным геномом, и других способов, рассматриваемые в настоящей заявке. Подходящие субъекты (например, пациенты) включают лабораторных животных (таких как мышь, крыса, кролик или морская свинка), сельскохозяйственных животных и домашних животных (таких как кошка или собака). Включены приматы, не являющиеся человеком и, предпочтительно, люди. Типичными субъектами являются пациенты-люди, у которых был диагностировано иммунное расстройство или есть риск его развития.
Используемый в настоящей заявке термин «пациент» относится к субъекту, у которого диагностировано иммунное расстройство, которое можно лечить с помощью вариантов нуклеазы, композиций для редактирования генома, векторов для генной терапии, векторов для редактирования генома, клеток с отредактированным геномом и других рассматриваемые в настоящей заявке способов.
Используемый в настоящей заявке термин «лечение» или «лечить» включает любое полезное или желательное воздействие на симптомы или патологию заболевания или патологического состояния и может включать даже минимальное снижение одного или нескольких измеримых маркеров заболевания или состояния, которое лечат, например, рака, БТПХ, инфекционных заболеваний, аутоиммунных заболеваний, воспалительных заболеваний и иммунодефицита. Лечение может включать задержку прогрессирования заболевания или состояния. «Лечение» не обязательно указывает на полное искоренение или излечение заболевания или состояния или ассоциированных с ним симптомов.
Используемые в настоящей заявке термины «предотвращать» и подобные слова, такие как «предотвращение», «предотвращенный», «профилактика» и т.д., указывают на подход к предотвращению, ингибированию или уменьшению вероятности возникновения или рецидива заболевания или состояния, например, рака, БТПХ, инфекционных заболеваний, аутоиммунных заболеваний, воспалительных заболеваний и иммунодефицита. Это также относится к задержке появления или рецидива заболевания или состояния или к задержке появления или повторения симптомов указанного заболевания или состояния. Используемый в настоящей заявке термин «предотвращение» и подобные слова также включает в себя снижение интенсивности, эффекта, симптомов и/или бремени заболевания или состояния до начала или рецидива указанного заболевания или состояния.
Используемое в настоящей заявке выражение «облегчение по меньшей мере одного симптома» относится к уменьшению одного или нескольких симптомов заболевания или состояния, от которого лечат субъекта, например, рака, БТПХ, инфекционного заболевания, аутоиммунного заболевания, воспалительного заболевания и иммунодефицита. В частных вариантах реализации заболеванием или состоянием, подвергаемым лечению, является рак, при котором один или более облегчаемых симптомов включают, среди прочего, слабость, усталость, одышку, склонность к кровоподтекам и кровотечению, частые инфекции, увеличенные лимфатические узлы, вздутый или болезненный живот (из-за увеличенных органов брюшной полости), боль в костях или суставах, переломы, незапланированную потерю веса, плохой аппетит, ночную потливость, постоянную легкую лихорадку и снижение мочеиспускания (из-за нарушения функции почек)).
Используемый в настоящей заявке термин «количество» относится к «количеству, эффективному» или «эффективному количеству» варианта нуклеазы, композиции для редактирования генома или клеток с отредактированным геномом, достаточному для достижения полезного или желаемого профилактического или терапевтического результата, включая клинические результаты.
«Профилактически эффективное количество» относится к количеству варианта нуклеазы, композиции для редактирования генома или клеток с отредактированным геномом, достаточному для достижения желаемого профилактического результата. Как правило, но не обязательно, так как профилактическая доза используется у субъектов до заболевания или на более ранней стадии заболевания, профилактически эффективное количество меньше терапевтически эффективного количества.
«Терапевтически эффективное количество» варианта нуклеазы, композиции для редактирования генома или клеток с отредактированным геномом может варьироваться в зависимости от таких факторов, как статус заболевания, возраст, пол и вес индивидуума, а также способность вызывать желаемый ответ у этого индивидуума. Терапевтически эффективным также является количество, при котором любые токсические или вредные эффекты перевешиваются терапевтически полезными эффектами. Термин «терапевтически эффективное количество» включает количество, которое эффективно для «лечения» субъекта (например, пациента). Когда указывается терапевтическое количество, точное количество композиций, рассматриваемых в частных вариантах реализации подлежащее введению, может быть определено врачом с учетом спецификации и с учетом индивидуальных различий в возрасте, весе, размере опухоли, степени инфекция или метастазирования, а также состояния пациента (субъекта).
C. ВАРИАНТЫ НУКЛЕАЗЫ
Варианты нуклеазы, рассматриваемые в настоящей заявке в частных вариантах реализации пригодны для геномного редактирования сайта-мишени в гене PD-1 и содержат один или более ДНК-связывающих доменов и один или более доменов расщепления ДНК (например, один или более доменов эндонуклеазы и/или экзонуклеазы) и, необязательно, один или более линкеров, рассматриваемых в настоящей заявке. Термины «перепрограммированная нуклеаза», «сконструированная нуклеаза» или «вариант нуклеазы» используются взаимозаменяемо и относятся к нуклеазе, содержащей один или более ДНК-связывающих доменов и один или более доменов расщепления ДНК, причем такая нуклеаза была разработана и/или модифицирована из родительской или встречающейся в природе нуклеазы, для связывания и расщепления двухцепочечной последовательности-мишени ДНК в гене PD-1.
В частных вариантах реализации вариант нуклеазы связывает и расщепляет последовательность-мишень в экзоне 5 гена PD-1, предпочтительно в SEQ ID NO: 25 в экзоне 5 гена PD-1 и более предпочтительно в последовательности «ATAC» в SEQ ID NO: 25 в экзоне 5 гена PD-1.
В частных вариантах реализации вариант нуклеазы связывает и расщепляет последовательность-мишень в экзоне 1 гена PD-1, предпочтительно с SEQ ID NO: 30 в экзоне 1 гена PD-1 и более предпочтительно в последовательности «ATCC» в SEQ ID NO: 30 в экзоне 1 гена PD-1.
В частных вариантах реализации вариант нуклеазы связывает и расщепляет последовательность-мишень в экзоне 2 гена PD-1, предпочтительно при SEQ ID NO: 35 в экзоне 2 гена PD-1 и более предпочтительно в последовательности «ACTT» в SEQ ID NO: 35 в экзоне 2 гена PD-1.
Такой вариант нуклеазы может быть сконструирован и/или модифицирован из встречающейся в природе нуклеазы или из предшествующего варианта нуклеазы. Варианты нуклеазы, рассматриваемые в частных вариантах реализации могут дополнительно содержать один или более дополнительных функциональных доменов, например, ферментативный домен конечного процессинга фермента, изменяющего структуру концов, который демонстрирует 5'-3'-экзонуклеазную, 5'-3'-щелочную экзонуклеазную, 3'-5'- экзонуклеазную (например, Trex2), 5'-flap-эндонуклеазную, геликазную, матрично-зависимую ДНК-полимеразную или матрично-независимую ДНК-полимеразнную активность.
Иллюстративные примеры вариантов нуклеаз, которые связывают и расщепляют последовательность-мишень в гене PD-1, включают, среди прочего, варианты хоминг-эндонуклеазы (мегануклеазы) и megaTAL.
1. Варианты хоминг-эндонуклеазы (мегануклеазы)
В различных вариантах реализации хоминг-эндонуклеазу или мегануклеазу перепрограммируют для введения двухцепочечного разрыва (DSB) в сайт-мишень в гене PD-1. В частных вариантах реализации вариант хоминг-эндонуклеазы вводит двухцепочечный разрыв в экзоне 5 гена PD-1, предпочтительно в SEQ ID NO: 25 в экзоне 5 гена PD-1 и более предпочтительно в последовательности «ATAC» в SEQ ID NO: 25 в экзоне 5 гена PD-1. В частных вариантах реализации вариант хоминг-эндонуклеазы вводит двухцепочечный разрыв в экзоне 1 гена PD-1, предпочтительно в SEQ ID NO: 30 в экзоне 1 гена PD-1 и более предпочтительно в последовательности «ATCC» в SEQ ID NO: 30 в экзоне 1 гена PD-1. В частных вариантах реализации вариант хоминг-эндонуклеазы вводит двухцепочечный разрыв в экзоне 2 гена PD-1, предпочтительно в SEQ ID NO: 35 в экзоне 2 гена PD-1 и более предпочтительно в последовательности «ACTT» в SEQ ID NO: 35 в экзоне 2 гена PD-1.
Термины «хоминг-эндонуклеаза» и «мегануклеаза» используются взаимозаменяемо и относятся к естественным хоминг-эндонуклеазам, которые распознают сайты расщепления 12-45 пар нуклеотидоа и обычно группируются в пять семейств на основе мотивов последовательности и структуры: LAGLIDADG, GIY-YIG HNH, бокс His-Cys и PD-(D/E)XK.
Термины «эталонная хоминг-эндонуклеаза» или «эталонная мегануклеаза» относятся к дикому типу хоминг-эндонуклеазы или хоминг-эндонуклеазе, встречающейся в природе. В одном варианте реализации «эталонная хоминг-эндонуклеаза» относится к хоминг-эндонуклеазе дикого типа, которая была модифицирована для увеличения базальной активности.
Термины «сконструированная хоминг-эндонуклеаза», «перепрограммированная хоминг-эндонуклеаза», «вариант хоминг-эндонуклеазы», «сконструированная мегануклеаза», «перепрограммированная мегануклеаза» или «вариант мегануклеазы» относятся к хоминг-эндонуклеазе, содержащей один или больше ДНК-связывающих доменов и один или более доменов расщепления ДНК, причем указанная хоминг-эндонуклеаза была сконструирована и/или модифицирована из родительской или природной хоминг-эндонуклеазы для связывания и расщепления последовательности-мишени ДНК в гене PD-1. Вариант хоминг-эндонуклеазы может быть сконструирован и/или модифицирован из природной хоминг-эндонуклеазы или из другого варианта хоминг-эндонуклеазы. Варианты хоминг-эндонуклеазы, рассматриваемые в частных вариантах реализации могут дополнительно содержать один или более дополнительных функциональных доменов, например, ферментативный домен конечного процессинга фермента, изменяющего структуру концов, который демонстрирует 5'-3'-экзонуклеазную, 5'-3'-щелочную экзонуклеазную, 3'-5'-экзонуклеазную (например, Trex2), 5'-flap-эндонуклеазную, геликазную, матрично-зависимую ДНК-полимеразную или матрично-независимую ДНК-полимеразнную активность.
Варианты хоминг-эндонуклеазы (HE) не существуют в природе и могут быть получены с помощью технологии рекомбинантной ДНК или случайного мутагенеза. Варианты HE могут быть получены путем внесения одного или нескольких аминокислотных изменений, например, мутации, замены, добавления или удаления одной или нескольких аминокислот во встречающихся в природе HE или варианте HE. В частных вариантах реализации вариант HE содержит одну или более аминокислотных замен в интерфейсе распознавания ДНК.
Варианты HE, рассматриваемые в частных вариантах реализации могут, кроме того, включать один или более линкеров и/или дополнительных функциональных доменов, например, ферментативный домен конечного процессинга фермента, изменяющего структуру концов, который демонстрирует 5'-3'-экзонуклеазную, 5'-3'-щелочную экзонуклеазную, 3'-5'- экзонуклеазную (например, Trex2), 5'-flap-эндонуклеазную, геликазную, матрично-зависимую ДНК-полимеразную или матрично-независимую ДНК-полимеразнную активность. В частных вариантах реализации варианты HE вводят в Т-клетку ферментом, изменяющим концы концов, который демонстрирует 5'-3'-экзонуклеазную, 5'-3'-щелочную экзонуклеазную, 3'-5'-экзонуклеазную (например, Trex2), 5'-flap-эндонуклеазную, геликазную, матрично-зависимую ДНК-полимеразную или матрично-независимую ДНК-полимеразнную активность. Вариант HE и фермент 3'-процессинга могут быть введены по отдельности, например, в разных векторах или в отдельных мРНК, или вместе, например, в виде слитого белка или в полицистронной конструкции, разделенной вирусным саморасщепляемым пептидом или элементом IRES.
Термины «интерфейс распознавания ДНК» относится к аминокислотным остаткам HE, которые взаимодействуют с основаниями-мишенями нуклеиновых кислот, а также к непосредственно примыкающим остаткам. Для каждой НЕ интерфейс распознавания ДНК включает в себя обширную сеть контактов боковая цепь-боковая цепь и боковая цепь-ДНК, большинство из которых обязательно уникальны для распознавания конкретной последовательности-мишени нуклеиновой кислоты. Таким образом, аминокислотная последовательность интерфейса распознавания ДНК, соответствующая конкретной последовательности нуклеиновой кислоты, значительно варьируется и является признаком любого природного варианта или варианта HE. В качестве неограничивающего примера, вариант HE, рассматриваемый в частных вариантах реализации может быть получен путем конструирования библиотек вариантов HE, в которых один или более аминокислотных остатков, локализованных в интерфейсе распознавания ДНК природной HE (или ранее созданного варианта HE), варьируются. Библиотеки могут быть подвергнуты скринингу на целевую активность расщепления против каждой предсказанного сайта-мишени PD-1 с помощью анализов расщепления (см., например, Jarjour et al., 2009. Nuc. Acids Res. 37(20): 6871-6880).
Хоминг-эндонуклеазы LAGLIDADG (LHE) являются наиболее хорошо изученным семейством хоминг-эндонуклеаз, кодируются в основном у архей и в органеллярной ДНК зеленых водорослей и грибов и обладают самой высокой общей специфичностью распознавания ДНК. LHE содержат один или два каталитических мотива LAGLIDADG на цепочку белка и функционируют как гомодимеры или одноцепочечные мономеры соответственно. Структурные исследования белков LAGLIDADG выявили высококонсервативную структуру ядра (Stoddard 2005), характеризующуюся складкой αββαββα, причем мотив LAGLIDADG принадлежит первой спирали указанной складки. Высокоэффективное и специфичное расщепление LHE представляет собой каркас белка для получения новых высокоспецифичных эндонуклеаз. Однако генное конструирование LHE для связывания и расщепления неприродного или неканонического сайта-мишени требуют выбора подходящего каркаса LHE, исследования локуса-мишени, выбора предполагаемых сайтов-мишеней и обширного изменения LHE для изменения точек контакта. и специфичности расщепления ее ДНК, до двух третей позиций пар нуклеотидов в сайте-мишени.
В одном варианте реализации LHE, из которых могут быть сконструированы перепрограммированные LHE или варианты LHE, включают, среди прочего, I-CreI и I-SceI.
Иллюстративные примеры LHE, из которых могут быть сконструированы перепрограммированные LHE или варианты LHE, включают, среди прочего, I-AabMI, I-AaeMI, I-AniI, I-ApaMI, I-CapIII, I-CapIV, I-CkaMI, I-CpaMI, I-CpaMII, I-CpaMIII, I-CpaMIV, I-CpaMV, I-CpaV, I-CraMI, I-EjeMI, I-GpeMI, I-GpiI, I-GzeMI, I-GzeMII, I-GzeMIII, I-HjeMI, I-LtrII, I-LtrI, I-LtrWI, I-MpeMI, I-MveMI, I-NcrII, I-Ncrl, I-NcrMI, I-OheMI, I-OnuI, I-OsoMI, I-OsoMII, I-OsoMIII, I-OsoMIV, I-PanMI, I-PanMII, I-PanMIII, I-PnoMI, I-ScuMI, I-SmaMI, I-SscMI и I-Vdi141I.
В одном варианте реализации перепрограммированная LHE или вариант LHE выбраны из группы, состоящей из: варианта I-CpaMI, варианта I-HjeMI, варианта I-OnuI, варианта I-PanMI и варианта I-SmaMI.
В одном варианте реализации перепрограммированная LHE или вариант LHE представляет собой вариант I-OnuI. См., например, SEQ ID NO: 6-14 и 60-63.
В одном варианте реализации перепрограммированные LHE I-OnuI или варианты I-OnuI, нацеленные на ген PD-1, были сконструированы из природного I-OnuI или его биологически активного фрагмента (SEQ ID NO: 1-5). В предпочтительном варианте реализации перепрограммированные LHE I-OnuI или варианты I-OnuI, нацеленные на ген PD-1 человека, были сконструированы из существующего варианта I-OnuI. В одном варианте реализации, перепрограммированные LHE I-OnuI были сконструированы против сайта-мишени гена PD-1 человека, указанного в SEQ ID NO: 25. В одном варианте реализации, перепрограммированные LHE I-OnuI OnuI были сконструированы против сайта-мишени гена PD-1 человека, указанного в SEQ ID NO: 30. В одном варианте реализации перепрограммированные I-OnuI LHE были сконструированы против сайта-мишени гена PD-1 человека, указанного в SEQ ID NO: 35.
В частном варианте реализации перепрограммированные I-OnuI LHE или вариант I-OnuI, который связывает и расщепляет ген PD-1 человека, содержит одну или более аминокислотных замен в интерфейсе распознавания ДНК. В частных вариантах реализацииI-OnuI LHE, который связывает и расщепляет ген PD-1 человека, содержит последовательность по меньшей мере на 70% по меньшей мере на 71%, по меньшей мере на 72%, по меньшей мере на 73%, по меньшей мере на 74%, по меньшей мере на 75%, по меньшей мере на 76%, по меньшей мере на 77%, по меньшей мере на 78%, по меньшей мере на 79%, по меньшей мере на 80%, по меньшей мере на 81%, по меньшей мере на 82%, по меньшей мере на 83%, по меньшей мере 8 на 4%, по меньшей мере на 85%, по меньшей мере на 86%, по меньшей мере на 87%, по меньшей мере на 88%, по меньшей мере на 89%, по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95% по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичную последовательности интерфейса распознавания ДНК I-OnuI (Taekuchi et al. 2011. Proc Natl Acad Sci USA 2011 Aug 9; 108 (32): 13077-13082) или варианта I-OnuI LHE, указанных в любой из SEQ ID NO: 6-14 и 60-63, их биологически активные фрагменты и/или их дополнительные варианты.
В одном варианте реализации I-OnuI LHE, который связывает и расщепляет ген PD-1 человека, содержит последовательность, по меньшей мере на 70%, более предпочтительно, по меньшей мере на 80%, более предпочтительно, по меньшей мере на 85%, более предпочтительно, по меньшей мере на 90%, более предпочтительно, по меньшей мере на 95%, более предпочтительно, по меньшей мере на 97%, более предпочтительно, по меньшей мере на 99% идентичную последовательности области контакта распознавания ДНК I-OnuI (Taekuchi et al. 2011. Proc Natl Acad Sci U. S. A. 2011 Aug 9; 108(32): 13077-13082) или варианта I-OnuI LHE, указанных в любой из SEQ ID NO: 6-14 и 60-63, их биологически активные фрагменты и/или их дополнительные варианты.
В частном варианте реализации вариант LHE I-OnuI, который связывает и расщепляет ген PD-1 человека, содержит одну или более аминокислотных замен или модификаций в интерфейсе распознавания ДНК I-OnuI, указанных в любой из SEQ ID NO: 1-14 и 60-63, их биологически активные фрагменты и/или их дополнительные варианты.
В частном варианте реализации вариант LHE I-OnuI, который связывает и расщепляет ген PD-1 человека, содержит одну или более аминокислотных замен или модификаций в интерфейсе распознавания ДНК, в частности в субмотивах, расположенных в позициях 24-50, 68. 82, 180-203 и 223-240 I-OnuI (SEQ ID NO: 1-5) или вариант I-OnuI, указанный в любой из SEQ ID NO: 6-14 и 60-63, их биологически активные фрагменты и/или их дополнительные варианты.
В частном варианте реализации I-OnuI LHE, который связывает и расщепляет ген PD-1 человека, содержит одну или более аминокислотных замен или модификаций в интерфейсе распознавания ДНК в положениях аминокислот, выбранных из группы, состоящей из: 19, 24, 26, 28, 30, 32, 34, 35, 36, 37, 38, 40, 42, 44, 46, 48, 68, 70, 72, 75, 76 77, 78, 80, 82, 168, 180, 182, 184, 186, 188, 189, 190, 191, 192, 193, 195, 197, 199, 201, 203, 223, 225, 227, 229, 231, 232, 234, 236, 238 и 240 в I-OnuI (SEQ ID NO: 1-5) или вариант I-OnuI, указанный в любой из SEQ ID NO: 6-14 и 60-63, их биологически активные фрагменты и/или их дополнительные варианты.
В частном варианте реализации вариант LHE I-OnuI, который связывает и расщепляет ген PD-1 человека, содержит 5, 10, 15, 20, 25, 30, 35 или 40 или более аминокислотных замен или модификаций в интерфейсе распознавания ДНК. в частности, в субмотивах, расположенных в положениях 24-50, 68-82, 180-203 и 223-240 I-OnuI (SEQ ID NO: 1-5) или вариант I-OnuI, указанный в любой из SEQ ID №: 6-14 и 60-63, их биологически активные фрагменты и/или их дополнительные варианты.
В частном варианте реализации вариант LHE I-OnuI, который связывает и расщепляет ген PD-1 человека, содержит 5, 10, 15, 20, 25, 30, 35 или 40 или более аминокислотных замен или модификаций в интерфейсе распознавания ДНК. В положениях аминокислот, выбранных из группы, состоящей из: 19, 24, 26, 28, 30, 32, 34, 35, 36, 37, 38, 40, 42, 44, 46, 48, 59, 68, 70, 72, 75, 76 77, 78, 80, 82, 168, 180, 182, 184, 186, 188, 189, 190, 191, 192, 193, 195, 197, 199, 201, 203, 223, 225, 227, 229, 231, 232, 234, 236, 238 и 240 в I-OnuI (SEQ ID NO: 1-5) или вариант I-OnuI, указанный в любой из SEQ ID NO: 6-14 и 60-63, их биологически активные фрагменты и/или их дополнительные варианты.
В одном варианте реализации вариант LHE I-OnuI, который связывает и расщепляет ген PD-1 человека, содержит одну или более аминокислотных замен или модификаций в дополнительных положениях, расположенных где-либо в пределах всей последовательности I-OnuI. Остатки, которые могут быть замещены и/или модифицированы, включают, среди прочего, аминокислоты, которые связываются с нуклеиновой кислотой-мишенью или которые взаимодействуют с основной цепью нуклеиновой кислоты или с нуклеотидными основаниями, непосредственно или через молекулу воды. В одном неограничивающем примере рассматриваемый в настоящей заявке вариант LHE I-OnuI, который связывает и расщепляет ген PD-1 человека, содержит одну или более замен и/или модификаций, предпочтительно по меньшей мере 5, предпочтительно по меньшей мере 10, предпочтительно по меньшей мере 15, предпочтительно по меньшей мере 20, более предпочтительно по меньшей мере 25, более предпочтительно по меньшей мере 30, еще более предпочтительно по меньшей мере 35 или даже более предпочтительно по меньшей мере 40 или более аминокислотных замен по меньшей мере в одном положении, выбранном из группы положений, состоящей из положений: 26, 28, 32, 34, 35, 36, 37, 38, 40, 42, 44, 46, 48, 59, 68, 70, 72, 75, 76, 78, 80, 138, 143, 159, 168, 178, 180, 182, 184, 186, 188, 189, 190, 191, 192, 193, 197, 199, 201, 203, 207, 223, 224, 225, 227, 229, 232, 236 и 238 в I-OnuI (SEQ ID NO: 1-5) или вариант I-OnuI, указанный в любой из SEQ ID NO: 6-14 и 60-63, их биологически активные фрагменты и/или их дополнительные варианты.
В некоторых вариантах реализации вариант HE расщепляет сайт-мишень экзона 5 PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно, по меньшей мере 25, более предпочтительно, по меньшей мере 35 или еще более предпочтительно, по меньшей мере 40 или более из следующих аминокислотных замен: L26Q, R28E, N32V, K34P, S35N, S36I, V37P, G38R, S40K, S40R, E42R, G44R, Q46E, T48D, N59D, V68K, A70Y, S72Q, S72R, N75S, A76Y, S78K, K80R, L138M, T143N, S159P, F168L, E178D, C180H, F182G, N184I, I186A, S188R, K189R, S190P, K191A, L192S, G193P, Q197R, V199R, S201A, S201M, T203G, K207R, Y223R, I224T, K225R, K229I, F232K, D236E и V238E в I-OnuI (SEQ ID NO: 1-5) или вариант I-OnuI, к указанный в любой из SEQ ID NO: 6-10, их биологически активные фрагменты и/или их дополнительные варианты.
В некоторых вариантах реализации вариант HE расщепляет сайт-мишень экзона 5 PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно, по меньшей мере 25, более предпочтительно, по меньшей мере 35 или даже более предпочтительно, по меньшей мере 40 или более из следующих аминокислотных замен: L26Q, R28E, N32V, K34P, S35N, S36I, V37P, G38R, S40K, E42R, G44R, Q46E, T48D, V68K, A70Y, S72Q, N75S, A76Y, S78K, K80R, L138M, T143N, S159P, F168L, C180H, F182G, N184I, I186A, S188R, K189R, S190P, K191A, L192S, G193P, Q197R, V199R, S201A, T203G, K207R, Y223R, K225R, K229I, F232K, D236E и V238E в I-OnuI (SEQ ID NO: 1-5) или вариант I-OnuI, указанный в любой из SEQ ID NO: 6-10, их биологически активные фрагменты и/или их дополнительные варианты.
В частных вариантах реализации вариант HE расщепляет сайт-мишень экзона 5 PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно, по меньшей мере 25, более предпочтительно, по меньшей мере 35 или еще более предпочтительно, по меньшей мере 40 или более из следующих аминокислотных замен: L26Q, R28E, N32V, K34P, S35N, S36I, V37P, G38R, S40R, E42R, G44R, Q46E, T48D, N59D, V68K, A70Y, S72R, N75S, A76Y, K80R, L138M, T143N, S159P, F168L, C180H, F182G, N184I, I186A, S188R, K189R, S190P, K191A, L192S, G193P, Q197R, V199R, S201A, T203G, K207R, Y223R, I224T, K225R, K229I, F232K, D236E и V238E в I-OnuI (SEQ ID NO: 1-5) или вариант I-OnuI, указанный в любой из SEQ ID NO: 6-10, их биологически активные фрагменты и/или их дополнительные варианты.
В дополнительных вариантах реализации, вариант HE расщепляет сайт-мишень экзона 5 PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно по меньшей мере 25, более предпочтительно по меньшей мере 35 или еще более предпочтительно по меньшей мере 40 или более из следующих аминокислотных замен: L26Q, R28E, N32V, K34P, S35N, S36I, V37P, G38R, S40R, E42R, G44R, Q46E, T48D, N59D, V68K, A70Y, S72Q, N75S, A76Y, K80R, L138M, T143N, S159P, F168L, E178D, C180H, F182G, N184I, I186A, S188R, K189R, S190P, K191A, L192S, G193P, Q197R, V199R, S201A, T203G, K207R, Y223R, I224T, K225R, K229I, F232K, D236E и V238E в I-OnuI (SEQ ID NO: 1-5) или вариант I-OnuI, указанный в любой из SEQ ID NO: 6-10, их биологически активные фрагменты и/или их дополнительные варианты.
В других вариантах реализации, вариант HE расщепляет сайт-мишень экзона 5 PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно по меньшей мере 25, более предпочтительно по меньшей мере 35 или еще более предпочтительно по меньшей мере 40 или более из следующих аминокислотных замен: L26Q, R28E, N32V, K34P, S35N, S36I, V37P, G38R, S40R, E42R, G44R, Q46E, T48D, N59D, V68K, A70Y, S72Q, N75S, A76Y, K80R, L138M, T143N, S159P, F168L, E178D, C180H, F182G, N184I, I186A, S188R, K189R, S190P, K191A, L192S, G193P, Q197R, V199R, S201A, T203G, K207R, Y223R, K225R, F232K, D236E и V238E в I-OnuI (SEQ ID NO: 1-5) или вариант I-OnuI, указанный в любой из SEQ ID NO: 6-10, их биологически активные фрагменты и/или их дополнительные варианты.
В частных вариантах реализации вариант HE расщепляет сайт-мишень экзона 5 PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно по меньшей мере 25, более предпочтительно по меньшей мере 35 или еще более предпочтительно по меньшей мере 40 или более из следующих аминокислотных замен: L26Q, R28E, N32V, K34P, S35N, S36I, V37P, G38R, S40R, E42R, G44R, Q46E, T48D, N59D, V68K, A70Y, S72Q, N75S, A76Y, K80R, L138M, T143N, S159P, F168L, E178D, C180H, F182G, N184I, I186A, S188R, K189R, S190P, K191A, L192S, G193P, Q197R, V199R, S201M, T203G, Y223R, K225R, F232K, D236E и V238E в I-OnuI (SEQ ID NO: 1-5) или вариант I-OnuI, указанный в любой из SEQ ID NO: 6-10, их биологически активные фрагменты и/или их дополнительные варианты.
В частных вариантах реализации вариант HE расщепляет сайт-мишень экзона 1 PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно, по меньшей мере 25, более предпочтительно, по меньшей мере 35 или еще более предпочтительно, по меньшей мере 40 или более аминокислотных замен по меньшей мере в одной позиций, выбранной из группы позиций, состоящей из позиций: 26, 28, 30, 32, 34, 35, 36, 37, 38, 40, 42, 44, 46, 48, 68, 70, 72, 75, 76, 78, 80, 100, 132, 138, 143, 155, 159, 178, 180, 184, 186, 189, 190, 191, 192, 193, 195, 201, 203, 207, 223, 225, 227, 232, 236, 238 и 240 в I-OnuI (SEQ ID NO: 1-5) или вариант I-OnuI, указанный в любой из SEQ ID NO: 11-12 и 60-63, их биологически активные фрагменты и/или их дополнительные варианты.
В некоторых вариантах реализации вариант HE расщепляет сайт-мишень экзона 1 гена PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно по меньшей мере 25, более предпочтительно по меньшей мере 35 или еще более предпочтительно по меньшей мере 40 или более из следующих аминокислотных замен: L26G, R28S, R30L, N32R, K34R, S35G, S36T, V37A, G38R, S40H, E42R, G44S, Q46A, Q46T, T48V, V68I, A70T, S72D, N75R, A76Y, S78R, K80R, K80C, I100V, V132A, L138M, T143N, S155G, S159P, E178D, C180S, N184R, I186R, K189N, S190V, K191N, L192A, G193R, Q195R, S201E, T203S, K207R, Y223H, K225Y, K227G, F232R, D236Q, V238R и T240E в I-OnuI (SEQ ID NO: 1-5) или вариант I-OnuI, указанный в любой из SEQ ID NO: 11-12 и 60-63, их биологически активные фрагменты и/или их дополнительные варианты.
В других вариантах реализации, вариант HE расщепляет сайт-мишень экзона 1 гена PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно по меньшей мере 25, более предпочтительно по меньшей мере 35 или еще более предпочтительно по меньшей мере 40 или более из следующих аминокислотных замен: L26G, R28S, R30L, N32R, K34R, S35G, S36T, V37A, G38R, S40H, E42R, G44S, Q46A, T48V, V68I, A70T, S72D, N75R, A76Y, S78R, K80R, I100V, L138M, T143N, S159P, E178D, C180S, N184R, I186R, K189N, S190V, K191N, L192A, G193R, Q195R, S201E, T203S, K207R, Y223H, K225Y, K227G, F232R, D236Q, V238R и T240E в I-OnuI (SEQ ID NO: 1-5) или вариант I-OnuI, указанный в любой из SEQ ID NO: 11-12 и 60-63, их биологически активные фрагменты и/или их дополнительные варианты.
В некоторых вариантах реализации вариант HE расщепляет сайт-мишень экзона 1 гена PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно по меньшей мере 25, более предпочтительно по меньшей мере 35 или еще более предпочтительно по меньшей мере 40 или более из следующих аминокислотных замен: L26G, R28S, R30L, N32R, K34R, S35G, S36T, V37A, G38R, S40H, E42R, G44S, Q46T, T48V, V68I, A70T, S72D, N75R, A76Y, S78R, K80C, I100V, V132A, L138M, T143N, S155G, S159P, E178D, C180S, N184R, I186R, K189N, S190V, K191N, L192A, G193R, Q195R, S201E, T203S, K207R, Y223H, K225Y, K227G, F232R, D236Q, V238R и T240E в I-OnuI (SEQ ID NO: 1-5) или вариант I-OnuI, указанный в любой из SEQ ID NO: 11-12 и 60-63, их биологически активные фрагменты и/или их дополнительные варианты.
В некоторых вариантах реализации вариант HE расщепляет сайт-мишень экзона 1 гена PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно по меньшей мере 25, более предпочтительно по меньшей мере 35 или еще более предпочтительно по меньшей мере 40 или более из следующих аминокислотных замен: L26G, R28S, R30L, N32R, K34R, S35G, S36T, V37A, G38R, S40H, E42R, G44S, Q46T, T48M, V68I, A70T, S72N, N75H, A76Y, S78T, K80R, I100V, L138M, T143N, S159P, E178D, C180S, N184R, I186R, K189N, S190V, K191N, L192A, G193R, Q195R, S201E, T203S, K207R, Y223H, K225Y, K227G, F232R, D236Q, V238R и T240E в I-OnuI (SEQ ID NO: 1-5) или вариант I-OnuI, указанный в любой из SEQ ID NO: 11-12 и 60-63, их биологически активные фрагменты и/или их дополнительные варианты.
В некоторых вариантах реализации вариант HE расщепляет сайт-мишень экзона 1 гена PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно по меньшей мере 25, более предпочтительно по меньшей мере 35 или еще более предпочтительно по меньшей мере 40 или более из следующих аминокислотных замен: L26G, R28S, R30L, N32R, K34R, S35G, S36T, V37A, G38R, S40H, E42R, G44S, Q46T, T48M, V68S, A70L, S72N, N75H, A76Y, K80V, T82Y, L138M, T143N, S159P, E178D, C180S, N184R, I186R, K189N, S190V, K191N, L192A, G193R, Q195R, S201E, T203S, K207R, Y223H, K225Y, K227G, F232R, D236Q, V238R и T240E в I-OnuI (SEQ ID NO: 1-5) или вариант I-OnuI, указанный в любой из SEQ ID NO: 11-12 и 60-63, их биологически активные фрагменты и/или их дополнительные варианты.
В некоторых вариантах реализации вариант HE расщепляет сайт-мишень экзона 1 гена PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно по меньшей мере 25, более предпочтительно по меньшей мере 35 или еще более предпочтительно по меньшей мере 40 или более из следующих аминокислотных замен: L26G, R28S, R30L, N32R, K34R, S35G, S36T, V37G, G38R, S40H, E42R, G44S, Q46T, T48M, V68S, A70T, S72N, N75H, A76Y, K80V, T82Y, L138M, T143N, S159P, E178D, C180S, N184R, I186R, K189N, S190V, K191N, L192A, G193R, Q195R, S201E, T203S, K207R, Y223H, K225Y, K227G, F232R, D236Q, V238R и T240E в I-OnuI (SEQ ID NO: 1-5) или вариант I-OnuI, указанный в любой из SEQ ID NO: 11-12 и 60-63, их биологически активные фрагменты и/или их дополнительные варианты.
В некоторых вариантах реализации вариант HE расщепляет сайт-мишень экзона 1 гена PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно по меньшей мере 25, более предпочтительно по меньшей мере 35 или еще более предпочтительно по меньшей мере 40 или более из следующих аминокислотных замен: L26G, R28S, R30L, N32R, K34R, S35G, S36T, V37A, G38R, S40H, E42R, G44S, Q46T, T48M, V68S, A70Y, S72N, N75H, A76Y, K80E, T82F, L138M, T143N, S159P, E178D, C180S, N184R, I186R, K189N, S190V, K191N, L192A, G193R, Q195R, S201E, T203S, K207R, Y223H, K225Y, K227G, F232R, D236Q, V238R и T240E в I-OnuI (SEQ ID NO: 1-5) или вариант I-OnuI, указанный в любой из SEQ ID NO: 11-12 и 60-63, их биологически активные фрагменты и/или их дополнительные варианты.
В частных вариантах реализации вариант HE расщепляет сайт-мишень экзона 2 гена PD-1 и содержит по меньшей мере 5, по меньшей мере 15, предпочтительно, по меньшей мере 25, более предпочтительно, по меньшей мере 35 или даже более предпочтительно, по меньшей мере 40 или более аминокислотных замен по меньшей мере в одной позиция, выбранная из группы позиций, состоящей из позиций: 24, 26, 28, 30, 32, 34, 35, 36, 37, 38, 40, 41, 42, 44, 46, 48, 68, 70, 72, 74, 75, 76, 78, 80, 82, 116, 138, 143, 159, 168, 178, 180, 182, 184, 186, 188, 189, 190, 191, 192, 193, 195, 199, 203, 207, 225, 227, 229, 232, 236 и 238 в I-OnuI (SEQ ID NO: 1-5) или вариант I-OnuI, указанный в любой из SEQ ID NO: 13-14, их биологически активные фрагменты и/или их дополнительные варианты.
В дополнительных вариантах реализации, вариант HE содержит по меньшей мере 5, по меньшей мере 15, предпочтительно, по меньшей мере 25, более предпочтительно, по меньшей мере 35 или даже более предпочтительно, по меньшей мере 40 или более из следующих аминокислотных замен: S24C, L26Q, R28Y, R28H, R30S, N32V, N32L, K34N, K34R, S35N, S35T, S36R, V37S, V37T, G38R, G38K, S40R, T41A, E42R, G44S, G44R, Q46E, Q46A, T48E, V68I, A70N, S72I, D74N, N75T, N75R, A76S, A76R, S78R, K80S, T82G, T82R, V116L, L138M, T143N, S159P, F168L, E178D, C180N, F182Y, N184H, I186K, K189G, S190R, K191T, L192T, G193R, Q195Y, V199R, T203A, T203S, K207R, K225N, K225T, K227W, K227S, K229A, K229P, F232R, W234A, W234D, D236E и V238R в I-OnuI (SEQ ID NO: 1-5) или вариант I-OnuI, указанный в любой из SEQ ID NO: 13-14, их биологически активные фрагменты и/или их дополнительные варианты.
В частных вариантах реализации вариант HE содержит по меньшей мере 5, по меньшей мере 15, предпочтительно, по меньшей мере 25, более предпочтительно, по меньшей мере 35 или даже более предпочтительно, по меньшей мере 40 или более из следующих аминокислотных замен: L26Q, R28Y, R30S, N32V, K34N, S35N, S36R, V37S, G38R, S40R, T41A, E42R, G44R, Q46A, T48E, A70N, S72I, N75T, A76S, S78R, K80S, T82G, L138M, T143N, S159P, F168L, E178D, C180N, F182Y, N184H, I186K, K189G, S190R, K191T, L192T, G193R, Q195Y, V199R, T203A, K207R, K225N, K227W, K229A, F232R, W234A, D236E и V238R в I-OnuI (SEQ ID NO: 1-5) или вариант I-OnuI, указанный в любой из SEQ ID NO: 13-14, их биологически активные фрагменты и/или их дополнительные варианты.
В некоторых вариантах вариант HE содержит по меньшей мере 5, по меньшей мере 15, предпочтительно, по меньшей мере 25, более предпочтительно, по меньшей мере 35 или даже более предпочтительно, по меньшей мере 40 или более из следующих аминокислотных замен: S24C, R28H, N32L, K34R, S35T, V37T, G38K, S40R, E42R, G44S, Q46E, T48E, V68I, A70N, S72I, D74N, N75R, A76R, S78R, K80S, T82R, V116L, L138M, T143N, S159P, F168L, E178D, C180N, F182Y, N184H, I186K, K189G, S190R, K191T, L192T, G193R, Q195Y, V199R, T203S, K207R, K225T, K227S, K229P, F232R, W234D, D236E и V238R в I-OnuI (SEQ ID NO: 1-5) или вариант I-OnuI, указанный в любой из SEQ ID NO: 13-14, их биологически активные фрагменты и/или их дополнительные варианты.
В частных вариантах реализации вариант I-OnuI LHE, который связывает и расщепляет ген PD-1 человека, содержит аминокислотную последовательность, которая по меньшей мере на 80%, предпочтительно по меньшей мере на 85%, более предпочтительно по меньшей мере на 90% или даже более предпочтительно по меньшей мере на 95% идентична аминокислотной последовательности, указанной в любой из SEQ ID NO: 6-14 и 60-63, или соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант I-OnuI LHE содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 6-14 и 60-63, или соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант I-OnuI LHE содержит аминокислотную последовательность, представленную в SEQ ID NO: 6, или соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант I-OnuI LHE содержит аминокислотную последовательность, представленную в SEQ ID NO: 7, или соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант I-OnuI LHE содержит аминокислотную последовательность, представленную в SEQ ID NO: 8, или соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант I-OnuI LHE содержит аминокислотную последовательность, представленную в SEQ ID NO: 9, или соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант I-OnuI LHE содержит аминокислотную последовательность, представленную в SEQ ID NO: 10, или соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант I-OnuI LHE содержит аминокислотную последовательность, представленную в SEQ ID NO: 11, или соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант I-OnuI LHE содержит аминокислотную последовательность, представленную в SEQ ID NO: 12, или соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант I-OnuI LHE содержит аминокислотную последовательность, представленную в SEQ ID NO: 13, или соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант I-OnuI LHE содержит аминокислотную последовательность, представленную в SEQ ID NO: 14, или соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант I-OnuI LHE содержит аминокислотную последовательность, представленную в SEQ ID NO: 60, или соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант I-OnuI LHE содержит аминокислотную последовательность, представленную в SEQ ID NO: 61, или соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант I-OnuI LHE содержит аминокислотную последовательность, представленную в SEQ ID NO: 62, или соответствующий биологически активный фрагмент.
В частных вариантах реализации вариант I-OnuI LHE содержит аминокислотную последовательность, представленную в SEQ ID NO: 63, или соответствующий биологически активный фрагмент.
MegaTAL
В различных вариантах реализации megaTAL, содержащий вариант хоминг-эндонуклеазы, перепрограммирован для введения двухцепочечного разрыва (DSB) в сайт-мишень в гене PD-1. В частных вариантах реализации megaTAL вводит DSB в экзон 5 гена PD-1, предпочтительно в SEQ ID NO: 27 в экзон 5 гена PD-1 и более предпочтительно в последовательности «ATAC» в SEQ ID NO: 27 в экзон 5 гена PD-1. В частных вариантах реализации megaTAL вводит двухцепочечный разрыв в экзон 1 гена PD-1, предпочтительно в SEQ ID NO: 32 в экзон 1 гена PD-1, и более предпочтительно в последовательности «ATCC» в SEQ ID NO: 32 в экзон 1 гена PD-1. В частных вариантах реализации megaTAL вводит двухцепочечный разрыв в экзон 2 гена PD-1, предпочтительно в SEQ ID NO: 37 в экзон 2 гена PD-1 и более предпочтительно в последовательности «ACTT» в SEQ ID NO: 37 в экзон 2 гена PD-1.
Термин «megaTAL» относится к полипептиду, содержащему связывающий домен ДНК TALE и вариант хоминг-эндонуклеазы, который связывает и расщепляет последовательность-мишень ДНК в гене PD-1 и необязательно содержит один или более линкеров и/или дополнительных функциональных доменов, например, ферментативный домен конечного процессинга фермента, изменяющего структуру концов, который демонстрирует 5'-3'-экзонуклеазную, 5'-3'-щелочную экзонуклеазную, 3'-5'-экзонуклеазную (например, Trex2), 5'-flap-эндонуклеазную, геликазную, матрично-зависимую ДНК-полимеразную или матрично-независимую ДНК-полимеразную активность.
В частных вариантах реализации megaTAL может быть введен в клетку вместе с ферментом, изменяющий структуру концов, который демонстрирует 5'-3'-экзонуклеазную, 5'-3'-щелочную экзонуклеазную, 3'-5'-экзонуклеазную (например, Trex2), 5'-flap-эндонуклеазную, геликазную, матрично-зависимую ДНК-полимеразную или матрично-независимую ДНК-полимеразнную активность. MegaTAL и 3'-процессинговый фермент могут быть введены отдельно, например, в разных векторах или отдельных мРНК, или вместе, например, в виде слитого белок или в полицистронной конструкции, отделенной вирусным саморасщепляемым пептидом или элементом IRES.
«ДНК-связывающий домен TALE» - это ДНК-связывающая часть эффекторов, подобных активаторам транскрипции (TALE или TAL-эффекторы), которые имитируют транскрипционный активаторы растений для манипулирования транскриптомом растения (см., например, Kay et al., 2007. Science 318: 648-651). ДНК-связывающие домены TALE, рассматриваемые в частных вариантах реализации являются сконструированно сконструированными de novo или из естественных TALE, например, AvrBs3 из Xanthomonas campestris pv. vesicatoria, Xanthomonas gardneri, Xanthomonas translucens, Xanthomonas axonopodis, Xanthomonas perforans, Xanthomonas alfalfa, Xanthomonas citri, Xanthomonas euvesicatoria и Xanthomonas oryzae и brg11 и hpx17 из Ralstonia solanacearum. Иллюстративные примеры белков TALE для получения и конструирования ДНК-связывающих доменов раскрыты в патенте США №9019767 и цитированных в нем ссылках, все из которых включены в настоящее описание в качестве ссылки во всей их полноте.
В частных вариантах реализации megaTAL содержит ДНК-связывающий домен TALE, содержащий одну или более повторяющихся единиц, которые участвуют в связывании ДНК-связывающего домена TALE с соответствующей последовательностью ДНК-мишени. Одна «повторяющаяся единица» (также называемая «повтор») обычно имеет длину 33-35 аминокислот.Каждая повторяющаяся единица ДНК-связывающего домена TALE содержит 1 или 2 ДНК-связывающих остатка, составляющих вариабельный ди-остаток повторения (RVD), обычно в положениях 12 и/или 13 повтора. Естественный (канонический) код для распознавания ДНК этих ДНК-связывающих доменов TALE был определен таким образом, что последовательность HD в положениях 12 и 13 приводит к связыванию с цитозином (C), NG связывается с T, NI с A, NN связывается с G или A, и NG связывается с T. В некоторых случаях предусмотрены неканонические (нетипичные) RVD.
Иллюстративные примеры неканонических RVD, подходящих для использования в конкретных megaTAL, рассмотренных в частных вариантах реализации включают, среди прочего, HH, KH, NH, NK, NQ, RH, RN, SS, NN, SN, KN для распознавания гуанина (G); NI, KI, RI, HI, SI для распознавания аденина (A); NG, HG, KG, RG для распознавания тимина (T); RD, SD, HD, ND, KD, YG для распознавания цитозина (C); NV, HN для распознавания A или G; и H*, HA, KA, N*, NA, NC, NS, RA, S* для распознавания A или T или G или C, где (*) означает, что аминокислота в положении 13 отсутствует.Дополнительные иллюстративные примеры RVD, подходящих для использования, в частности, в megaTAL, рассматриваемых в частных вариантах реализации дополнительно включают в себя примеры, приведенные в патенте США №8614092, который включен посредством ссылки во всей своей полноте.
В частных вариантах реализации megaTAL, рассматриваемый в настоящей заявке, содержит ДНК-связывающий домен TALE, содержащий от 3 до 30 повторяющихся единиц. В некоторых случаях megaTAL состоит из 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 повторяющихся единиц ДНК-связывающего домена TALE. В предпочтительном варианте реализации megaTAL, рассматриваемый в настоящей заявке, содержит связывающий домен ДНК TALE, содержащий 5-15 повторяющихся единиц, более предпочтительно, 7-15 повторяющихся единиц, более предпочтительно, 9-15 повторяющихся единиц, и, более предпочтительно, 9, 10, 11, 12, 13, 14 или 15 повторяющихся единиц.
В частных вариантах реализации megaTAL, рассматриваемый в настоящей заявке, содержит ДНК-связывающий домен TALE, содержащий от 3 до 30 повторяющихся единиц, и дополнительную одиночную усеченную повторяющуюся единицу TALE, содержащую 20 аминокислот, расположенных на С-конце набора повторяющихся единиц TALE, т.е. дополнительную повторяющуюся единицу С-концевого пол-ДНК-связывающего домена TALE (аминокислоты от -20 до -1 С-кэп, раскрытые ниже в настоящей заявке). Таким образом, рассматриваемый в настоящей заявке megaTAL содержит ДНК-связывающий домен TALE, содержащий от 3,5 до 30,5 повторяющихся единиц. В некоторых случаях megaTAL содержит 3,5, 4,5, 5,5, 6,5, 7,5, 8,5, 9,5, 10,5, 11,5, 12,5, 13,5, 14,5, 15,5, 16,5, 17,5, 18,5, 19,5, 20,5, 21,5, 22,5, 23,5, 24,5, 25,5, 26,5, 27,5, 28,5, 29,5 или 30,5 повторяющихся единиц ДНК-связывающего домена TALE. В предпочтительном варианте реализации megaTAL, рассматриваемый в настоящей заявке, содержит ДНК-связывающий домен TALE, содержащий 5,5-15,5 повторяющихся единиц, более предпочтительно 7,5-15,5 повторяющихся единиц, более предпочтительно 9,5-15,5 повторяющихся единиц и более предпочтительно 9,5, 10,5, 11,5, 12,5, 13,5, 14,5 или 15,5 повторяющихся единиц.
В частных вариантах реализации megaTAL содержит эффекторную структуру TAL, содержащую полипептид «N-концевой домен (NTD)», один или более повторяющихся доменов/единиц TALE, полипептид «C-концевой домен (CTD)» и вариант хоминг-эндонуклеазы. В некоторых вариантах реализации NTD, повторы TALE и/или домены CTD принадлежат к одному и тому же биологическому виду. В других вариантах реализации один или более доменов NTD, TALE и/или CTD относятся к разным видам.
Используемый в настоящей заявке термин «полипептид N-концевого домена (NTD)» относится к последовательности, которая фланкирует N-концевой участок или фрагмент встречающегося в природе ДНК-связывающего домена TALE. Последовательность NTD, если она присутствует, может иметь любую длину, пока повторяющиеся единицы ДНК-связывающего домена TALE сохраняют способность связывать ДНК. В частных вариантах реализации полипептид NTD содержит по меньшей мере от 120 до по меньшей мере 140 или более аминокислот, N-концевых по отношению к ДНК-связывающему домену TALE (0 представляет собой аминокислоту 1 большинства N-концевых повторяющихся единиц). В частных вариантах реализации полипептид NTD содержит по меньшей мере приблизительно 120, 121, 122, 123, 124, 125, 126, 127, 128, 129, 130, 131, 132, 133, 134, 135, 136, 137, 138, 139 или по меньшей мере 140 аминокислот, N-концевых к ДНК-связывающему домену TALE. В одном варианте реализации megaTAL, рассматриваемый в настоящей заявке, содержит полипептид NTD по меньшей мере с приблизительно аминокислотами от+1 до+122 и по меньшей мере приблизительно от+1 до+137 белка TALE Xanthomonas (0 представляет собой аминокислоту 1 наиболее N-концевой повторяющейся единицы). В частных вариантах реализации полипептид NTD содержит по меньшей мере приблизительно 122, 123, 124, 125, 126, 127, 128, 129, 130, 131, 132, 133, 134, 135, 136 или 137 аминокислот, N-концевых по отношению к ДНК-связывающему домену TALE белка TALE Xanthomonas. В одном варианте реализации megaTAL, рассматриваемый в настоящей заявке, содержит полипептид NTD по меньшей мере с аминокислотами от+1 до+121 белка TALE Ralstonia (0 представляет собой аминокислоту 1 наиболее N-концевой повторяющейся единицы). В частных вариантах реализации полипептид NTD содержит по меньшей мере приблизительно 121, 122, 123, 124, 125, 126, 127, 128, 129, 130, 131, 132, 133, 134, 135, 136 или 137 аминокислот, N-концевых по отношению к ДНК-связывающему домену TALE белка TALE Ralstonia.
Используемый в настоящей заявке термин «полипептид С-концевого домена (CTD)» относится к последовательности, которая фланкирует С-концевой участок или фрагмент встречающегося в природе ДНК-связывающего домена TALE. Последовательность CTD, если она присутствует, может иметь любую длину до тех пор, пока повторяющиеся единицы TALE ДНК-связывающего домена сохраняют способность связывать ДНК. В частных вариантах реализации полипептид CTD содержит по меньшей мере от 20 до по меньшей мере 85 или более аминокислот, С-концевых по отношению к последнему полному повтору ДНК-связывающего домена TALE (первые 20 аминокислот представляют собой полу-повторяющуюся единицу, С-концевую к последней полной С-концевой повторяющейся единице). В частных вариантах реализации полипептид CTD содержит по меньшей мере приблизительно 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 443, 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64, 65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84, или по меньшей мере 85 аминокислот, С-концевых по отношению к последнему полному повтору ДНК-связывающего домена TALE. В одном варианте реализации megaTAL, рассматриваемый в настоящей заявке, содержит полипептид CTD, содержащий по меньшей мере аминокислоты приблизительно от -20 до -1 белка TALE Xanthomonas (-20 представляет собой аминокислоту 1 полу-повторяющейся единицы, С-концевую к последней полной С-концевой повторяющейся единице). В частных вариантах реализации полипептид CTD содержит по меньшей мере приблизительно 20, 19, 18, 17, 16, 15, 14, 13, 12, 11, 10, 9, 8, 7, 6, 5, 4, 3, 2, или 1 аминокислоту, С-концевых по отношению к последнему полному повтору ДНК-связывающего домена TALE белка Xanthomonas. В одном варианте реализации megaTAL, рассматриваемый в настоящей заявке, содержит полипептид CTD, содержащий по меньшей мере аминокислоты приблизительно от -20 до -1 белка TALE Ralstonia (-20 представляет собой аминокислоту 1 полу-повторяющейся единицы, С-концевую к последней полной С-концевой повторяющейся единице). В частных вариантах реализации полипептид CTD содержит по меньшей мере приблизительно 20, 19, 18, 17, 16, 15, 14, 13, 12, 11, 10, 9, 8, 7, 6, 5, 4, 3, 2, или 1 аминокислоту, С-концевую к последнему полному повтору ДНК-связывающего домена TALE белка TALE Ralstonia.
В частных вариантах реализации megaTAL, рассматриваемый в настоящей заявке, содержит слитый полипептид, содержащий ДНК-связывающий домен TALE, сконструированный для связывания последовательности-мишени, хоминг-эндонуклеазу, перепрограммированную для связывания и расщепления последовательности-мишени, и, необязательно, полипептид NTD и/или CTD, необязательно связанные друг с другом одним или несколькими линкерными полипептидами, рассматриваемыми в другом месте настоящей заявки. Без привязки к какой-либо конкретной теории, предполагается, что megaTAL, содержащий ДНК-связывающий домен TALE и, необязательно, полипептид NTD и/или CTD, слит с линкерным полипептидом, который затем слит с вариантом хоминг-эндонуклеазы. Таким образом, ДНК-связывающий домен TALE связывает последовательность-мишень ДНК, которая находится в пределах 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14 или 15 нуклеотидов от последовательности-мишени, связанной ДНК-связывающим доменом варианта хоминг-эндонуклеазы. Таким образом, рассматриваемые в настоящей заявке megaTAL увеличивают специфичность и эффективность редактирования генома.
В одном варианте реализации megaTAL содержит вариант хоминг-эндонуклеазы и ДНК-связывающий домен TALE, который связывает нуклеотидную последовательность, которая находится в пределах приблизительно 2, 3, 4, 5 или 6 нуклеотидов, предпочтительно, на 2 или 4 нуклеотида в направлении против хода транскрипции от сайта связывания перепрограммированной хоминг-эндонуклеазы.
В одном варианте реализации, megaTAL содержит вариант хоминг- эндонуклеазы и ДНК-связывающий домен TALE, который связывает нуклеотидную последовательность, указанную в SEQ ID NO: 26, которая находится на расстоянии 5 нуклеотидов в направлении против хода транскрипции нуклеотидной последовательности, связанной и расщепленной вариантом хоминг-эндонуклеазы (SEQ ID NO: 25). В предпочтительных вариантах реализации последовательность-мишень megaTAL представляет собой SEQ ID NO: 27.
В одном варианте реализации, megaTAL содержит вариант хоминг- эндонуклеазы и ДНК-связывающий домен TALE, который связывает нуклеотидную последовательность, указанную в SEQ ID NO: 31, которая находится на расстоянии 2 нуклеотида в направлении против транскрипции от нуклеотидной последовательности, связанной и расщепленной вариантом хоминг-эндонуклеазы (SEQ ID NO: 30). В предпочтительных вариантах реализации последовательность-мишень megaTAL представляет собой SEQ ID NO: 32.
В одном варианте реализации, megaTAL содержит вариант хоминг- эндонуклеазы и ДНК-связывающий домен TALE, который связывает нуклеотидную последовательность, указанную в SEQ ID NO: 36, которая находится на расстоянии 5 нуклеотидов в направлении против транскрипции от нуклеотидной последовательности, связанной с нуклеотидной последовательностью, связанной и расщепленной вариантом хоминг-эндонуклеазы (SEQ ID NO: 35). В предпочтительных вариантах реализации целевая последовательность megaTAL представляет собой SEQ ID NO: 37.
В частных вариантах реализации megaTAL, рассматриваемый в настоящей заявке, содержит одну или более повторяющихся единиц ДНК-связывающего TALE и вариант LHE, сконструированный или перепрограммированный из LHE, выбранной из группы, состоящей из: I-AabMI, I-AaeMI, I-AniI, I-ApaMI, I-CapIII, I-CapIV, I-CkaMI, I-CpaMI, I-CpaMII, I-CpaMIII, I-CpaMIV, I-CpaMV, I-CpaV, I-CraMI, I-EjeMI, I-GpeMI, I-GpiI, I-GzeMI, I-GzeMII, I-GzeMIII, I-HjeMI, I-LtrII, I-LtrI, I-LtrWI, I-MpeMI, I-MveMI, I-NcrII, I-Ncrl, I-NcrMI, I-OheMI, I-OnuI, I-OsoMI, I-OsoMII, I-OsoMIII, I-OsoMIV, I-PanMI, I-PanMII, I-PanMIII, I-PnoMI, I-ScuMI, I-SmaMI, I-SscMI, I-Vdi141I и их варианты или предпочтительно I-CpaMI, I-HjeMI, I-OnuI, I-PanMI, SmaMI и их варианты или, более предпочтительно, I-OnuI и его варианты.
В частных вариантах реализации megaTAL рассматриваемый в настоящей заявке, содержит NTD, одну или более повторяющихся единиц ДНК-связывающего TALE, CTD и вариант LHE, выбранный из группы, состоящей из: I-AabMI, I-AaeMI, I-AniI, I-ApaMI, I-CapIII, I-CapIV, I-CkaMI, I-CpaMI, I-CpaMII, I-CpaMIII, I-CpaMIV, I-CpaMV, I-CpaV, I-CraMI, I-EjeMI, I-GpeMI, I-GpiI, I-GzeMI, I-GzeMII, I-GzeMIII, I-HjeMI, I-LtrII, I-LtrI, I-LtrWI, I-MpeMI, I-MveMI, I-NcrII, I-Ncrl, I-NcrMI, I-OheMI, I-OnuI, I-OsoMI, I-OsoMII, I-OsoMIII, I-OsoMIV, I-PanMI, I-PanMII, I-PanMIII, I-PnoMI, I-ScuMI, I-SmaMI, I-SscMI, I-Vdi141I и их варианты или предпочтительно I-CpaMI, I-HjeMI, I-OnuI, I-PanMI, SmaMI и их варианты, или более предпочтительно I-OnuI и его варианты.
В частных вариантах реализации megaTAL, рассматриваемый в настоящей заявке, содержит NTD, от приблизительно 9,5 до приблизительно 15,5 повторяющихся единиц ДНК-связывающего TALE, и вариант LHE, выбранный из группы, состоящей из: I-AabMI, I-AaeMI, I-AniI, I-ApaMI, I-CapIII, I-CapIV, I-CkaMI, I-CpaMI, I-CpaMII, I-CpaMIII, I-CpaMIV, I-CpaMV, I-CpaV, I-CraMI, I-EjeMI, I-GpeMI, I-GpiI, I-GzeMI, I-GzeMII, I-GzeMIII, I-HjeMI, I-LtrII, I-LtrI, I-LtrWI, I-MpeMI, I-MveMI, I-NcrII, I-Ncrl, I-NcrMI, I-OheMI, I-OnuI, I-OsoMI, I-OsoMII, I-OsoMIII, I-OsoMIV, I-PanMI, I-PanMII, I-PanMIII, I-PnoMI, I-ScuMI, I-SmaMI, I-SscMI, I-Vdi141I и их варианты или предпочтительно I-CpaMI, I-HjeMI, I-OnuI, I-PanMI, SmaMI и их варианты, или более предпочтительно I-OnuI и его варианты.
В частных вариантах реализации MegaTAL, рассматриваемый в настоящей заявке, содержит NTD длиной от приблизительно 122 аминокислот до 137 аминокислот, приблизительно 9,5, приблизительно 10,5, приблизительно 11,5, приблизительно 12,5, приблизительно 13,5, приблизительно 14,5 или приблизительно 15,5 повторяющихся единиц связывания, CTD длиной от приблизительно 20 аминокислот до приблизительно 85 аминокислот и вариант LHE I-OnuI. В частных вариантах реализации любой из, два из или все из NTD, ДНК-связывающего домена и CTD могут быть сконструированы из одного и того же биологического вида или разных видов в любой подходящей комбинации.
В частных вариантах реализации megaTAL, рассматриваемый в настоящей заявке, содержит аминокислотную последовательность, указанную в любой из SEQ ID NO: 15-23 и 64.
В некоторых вариантах реализации megaTAL, рассматриваемый в настоящей заявке, закодирован последовательностью мРНК, представленной в любой из SEQ ID NO: 40-42 и 65-68.
В частных вариантах реализации рассматриваемый в настоящей заявке слитый белок megaTAL-Trex2 содержит аминокислотную последовательность, представленную в SEQ ID NO: 24.
В некоторых вариантах реализации megaTAL содержит ДНК-связывающий домен TALE, а вариант LHE I-OnuI связывает и расщепляет нуклеотидную последовательность, указанную в SEQ ID NO: 27. В частных вариантах реализации megaTAL, который связывает и расщепляет нуклеотидную последовательность, указанную в SEQ ID. NO: 27 содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 15-19.
В некоторых вариантах реализации megaTAL содержит ДНК-связывающий домен TALE, а вариант I-OnuI LHE связывает и расщепляет нуклеотидную последовательность, указанную в SEQ ID NO: 32. В частных вариантах реализации megaTAL, который связывает и расщепляет нуклеотидную последовательность, указанную в SEQ ID NO: 32, содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 20-21 и 64.
В некоторых вариантах реализации megaTAL содержит ДНК-связывающий домен TALE, а вариант LHE I-OnuI связывает и расщепляет нуклеотидную последовательность, указанную в SEQ ID NO: 37. В частных вариантах реализации megaTAL связывает и расщепляет нуклеотидную последовательность, указанную в SEQ ID. NO: 37, содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 22-23.
2. Ферменты, изменяющие структуру концов
Композиции и способы редактирования генома, рассматриваемые в частных вариантах реализации включают редактирование клеточных геномов с использованием варианта нуклеазы и одной или нескольких копий фермента, изменяющего структуру концов. В частных вариантах реализации один полинуклеотид кодирует вариант хоминг-эндонуклеазы и фермент, изменяющий структуру концов, разделенные линкером, саморасщепляемой пептидной последовательностью, например, последовательностью 2A, или последовательностью IRES. В частных вариантах реализации композиции для редактирования генома содержат полинуклеотид, кодирующий вариант нуклеазы, и отдельный полинуклеотид, кодирующий фермент, изменяющий структуру концов. В частных вариантах реализации композиции для редактирования генома содержат полинуклеотид, кодирующий фермент варианта хоминг-эндонуклеазы, изменяющий структуру концов одного слитого полипептида, в дополнение к тандемной копии фермента, изменяющего структуру концов, разделенные саморасщепляемым пептидом.
Термин «фермент, изменяющий структуру концов» относится к ферменту, который модифицирует открытые концы полинуклеотидной цепи. Полинуклеотид может представлять собой двухцепочечную ДНК (дцДНК), одноцепочечную ДНК (оцДНК), РНК, двухцепочечные гибриды ДНК и РНК и синтетическую ДНК (например, содержащие основания, отличные от A, C, G и T). Фермент, изменяющий положение концов, может модифицировать открытые концы полинуклеотидной цепи путем добавления одного или нескольких нуклеотидов, удаления одного или нескольких нуклеотидов, удаления или модификации фосфатной группы и/или удаления или модификации гидроксильной группы. Фермент, изменяющий расположение концов, может модифицировать концы на сайтах разреза эндонуклеазой или на концах, создаваемых другими химическими или механическими средствами, такими как сдвиг (например, посредством прохождения через иглу малого калибра, нагревание, обработка ультразвуком, обработка в барабане с мини-шариками и распыление), ионизирующее излучение, ультрафиолетовое излучение, кислородные радикалы, химический гидролиз и химиотерапевтические агенты.
В частных вариантах реализации композиции и способы редактирования генома, рассматриваемые в частных вариантах реализации включают редактирование клеточных геномов с использованием варианта хоминг-эндонуклеазы или megaTAL и фермента, изменяющего структуру концов ДНК.
Термин «фермент, изменяющий структуру концов ДНК» относится к ферменту, который модифицирует открытые концы ДНК. Фермент, изменяющий структуру концов ДНК, может модифицировать тупые концы или ступенчатые концы («липкие» 5'- или 3'-концы). Фермент ДНК, изменяющий структуру концов, может модифицировать одноцепочечную или двухцепочечную ДНК. Фермент, изменяющий структуру концов ДНК, может модифицировать концы на сайтах разреза эндонуклеазой или на концах, генерируемых другими химическими или механическими средствами, такими как сдвиг (например, сдвиг (например, посредством прохождения через иглу малого калибра, нагревание, обработка ультразвуком, обработка в барабане с мини-шариками и распыление), ионизирующее излучение, ультрафиолетовое излучение, кислородные радикалы, химический гидролиз и химиотерапевтические агенты. ДНК-фермент, изменяющий структуру концов, может модифицировать открытые концы ДНК, добавляя один или более нуклеотидов, удаляя один или более нуклеотидов, удаляя или модифицируя фосфатную группу и/или удаляя или модифицируя гидроксильную группу.
Иллюстративные примеры ферментов, изменяющих структуру концов ДНК, подходящих для применения в частных вариантах реализации, рассматриваемые в настоящей заявке, включают, среди прочего: 5'-3' экзонуклеазы, 5'-3' щелочные экзонуклеазы, 3'-5' экзонуклеазы, 5' flap-эндонуклеазы, геликазы, фосфатазы, гидролазы и матрично-независимые ДНК-полимеразы.
Другие иллюстративные примеры ферментов, изменяющих структуру концов ДНК, подходящих для применения в частных вариантах реализации, рассматриваемые в настоящей заявке, включают, среди прочего, Trex2, Trex1, Trex1 без трансмембранного домена, Apollo, Artemis, DNA2, Exo1, ExoT, ExoIII, Fen1, Fan1, MreII, Rad2, Rad9, TdT (терминальная дезоксинуклеотидилтрансфераза), PNKP, RecE, RecJ, RecQ, лямбда-экзонуклеаза, Sox, ДНК-полимераза Vaccinia, экзонуклеаза I, экзонуклеаза III, экзонуклеаза VII, NDK1, NDK5, NDK7, NDR, NDR7, NRK T7-экзонуклеаза Gene 6, белок интеграции вируса миелобластоза птиц (IN), Bloom, антарктическая фосфатаза, щелочная фосфатаза, полинуклеотидкиназа (PNK), ApeI, нуклеаза бобов мунг, Hex1, TTRAP (TDP2), Sgs1, Sae2, CUP, Pol мю, Pol лямбда, MUS81, EME1, EME2, SLX1, SLX4 и UL-12.
В частных вариантах реализации композиции для редактирования генома и способы редактирования клеточных геномов, рассматриваемые в настоящей заявке, включают полипептиды, содержащие вариант хоминг-эндонуклеазы или megaTAL и экзонуклеазу. Термин «экзонуклеаза» относится к ферментам, которые расщепляют фосфодиэфирные связи на конце полинуклеотидной цепи посредством реакции гидролиза, разрушающей фосфодиэфирные связи на 3' или 5' конце.
Иллюстративные примеры экзонуклеаз, подходящих для применения в частных вариантах реализации, рассматриваемых в настоящей заявке, включают, среди прочего: hExoI, ExoI дрожжей, E.coli ExoI, hTREX2, TREX2 мыши, крысиный TREX2, hTREX1, TREX1 мыши, TREX1 крысы и Rat TREX1.
В частных вариантах реализации ДНК-фермент, изменяющий структуру концов, представляет собой экзонуклеазу от 3' до 5', предпочтительно Trex 1 или Trex2, более предпочтительно Trex2 и еще более предпочтительно Trex2 человека или мыши.
B. Сайты-мишени
Варианты нуклеазы, рассматриваемые в частных вариантах реализации могут быть предназначены для связывания с любой подходящей последовательностью-мишенью и могут иметь новую специфичность связывания по сравнению с природной нуклеазой. В частных вариантах реализации сайт-мишень представляет собой регуляторную область гена, включая, среди прочего, промоторы, энхансеры, репрессорные элементы и тому подобное. В частных вариантах реализации сайт-мишень представляет собой кодирующую область гена или сайт сплайсинга. В некоторых вариантах реализации нуклеазные варианты предназначены для подавления или снижения экспрессии гена. В частных вариантах реализации нуклеазный вариант и донорная матрица репарации могут быть предназначены для репарации или удаления желаемой целевой последовательности.
В различных вариантах варианты нуклеазы связывают и расщепляют последовательность-мишень в гене рецептора запрограммированной смерти 1 (PD-1). PD-1 также упоминается как рецептора запрограммированной смерти клеток 1 (PDCD1), восприимчивости к системной красной волчанке 2 (SLEB2), CD279, HPD1, PD1, HPD-L и HSLE1. PD-1 является членом семейства костимулирующих рецепторов B7/CD28. Молекула PD-1 состоит из внеклеточного лигандсвязывающего домена IgV, трансмембранного домена и внутриклеточного домена, который имеет потенциальные сайты фосфорилирования, расположенные с ингибирующим мотивом на основе иммунного тирозина (ITIM) и ингибирующим мотивом на основе иммунорецепторного тирозинового ингибирующего рецептора (ITSM). PD-1 представляет собой ингибирующий корецептор, экспрессируемый на Т-клетках, Tregs, истощенных Т-клетках, В-клетках, активированных моноцитах, дендритных клетках (DC), клетках естественных киллеров (NK) и клетках естественных киллерах T (NKT). PD-1 отрицательно регулирует активацию Т-клеток посредством связывания с его лигандами, запрограммированным лигандом смерти 1 (PD-L1) и запрограммированным лигандом смерти 2 (PD-L2). Связывание PD-1 ингибирует пролиферацию Т-клеток и выработку интерферона-γ (IFN-γ), фактора некроза опухоли-α и IL-2 и снижает выживаемость Т-клеток. Экспрессия PD-1 является отличительной чертой «истощенных» Т-клеток, которые испытали высокий уровень стимуляции. Указанное состояние истощения, которое возникает во время хронических инфекций и рака, характеризуется дисфункцией Т-клеток, что приводит к недостаточному контролю инфекций и опухолей.
В частных вариантах реализации вариант хоминг-эндонуклеазы или megaTAL вводит двухцепочечный разрыв (DSB) в сайт-мишень в гене PD-1. В частных вариантах реализации вариант хоминг-эндонуклеазы или megaTAL вводит DSB в экзоне 5 гена PD-1, предпочтительно в SEQ ID NO: 25 (или SEQ ID NO: 27) в экзоне 5 гена PD-1, и более предпочтительно в последовательности «ATAC» в SEQ ID NO: 25 (или SEQ ID NO: 27) в экзоне 5 гена PD-1.
В предпочтительном варианте реализации вариант хоминг-эндонуклеазы или megaTAL расщепляет двухцепочечную ДНК и вводит DSB в полинуклеотидную последовательность, представленную в SEQ ID NO: 25 или 27.
В частных вариантах реализации вариант хоминг-эндонуклеазы или megaTAL вводит DSB в экзоне 1 гена PD-1, предпочтительно в SEQ ID NO: 30 (или SEQ ID NO: 32) в экзоне 1 гена PD-1, и более предпочтительно в последовательности «ATCC» в SEQ ID NO: 30 (или SEQ ID NO: 32) в экзоне 1 гена PD-1.
В предпочтительном варианте реализации вариант хоминг-эндонуклеазы или megaTAL расщепляет двухцепочечную ДНК и вводит DSB в полинуклеотидную последовательность, представленную в SEQ ID NO: 30 или 32.
В частных вариантах реализации вариант хоминг-эндонуклеазы или megaTAL вводит DSB в экзоне 2 гена PD-1, предпочтительно в SEQ ID NO: 35 (или SEQ ID NO: 37) в экзоне 2 гена PD-1, и более предпочтительно в последовательности «ACTT» в SEQ ID NO: 35 (или SEQ ID NO: 37) в экзоне 2 гена PD-1.
В предпочтительном варианте реализации вариант хоминг-эндонуклеазы или megaTAL расщепляет двухцепочечную ДНК и вводит DSB в полинуклеотидную последовательность, представленную в SEQ ID NO: 35 или 37.
В предпочтительном варианте реализации ген PD-1 представляет собой ген PD-1 человека.
C. Донорные матрицы репарации
Для введения DSB в последовательность-мишень могут быть использованы варианты нуклеазы; DSB может быть репарирован с помощью механизмов гомологичной репарации (HDR) в присутствии одного или нескольких донорной матрицы репарации.
В различных вариантах реализации донорная матрица репарации содержит один или более полинуклеотидов, кодирующих энхансер иммунопотенции, демпфер иммуносупрессивного сигнала или сконструированный рецептор антигена.
В различных вариантах реализации предполагается, что введение в клетку сконструированной нуклеазы в присутствии множества донорных матриц репарации, независимо кодирующих энхансеры иммунопотенции и/или демпферы иммуносупрессивного сигнала, нацеленные на различные иммуносупрессивные пути, дает Т-клетки с отредактированным геномом с повышенной терапевтической эффективностью и жизнестойкостью. Например, в частных вариантах реализации изобретения предпочтительными могут быть энхансеры иммунопотенции или демпферы иммуносупрессивного сигнала, нацеленные на комбинации путей PD-1, LAG-3, CTLA-4, TIM3, IL-10R, TIGIT и TGFβRII.
В частных вариантах реализации донорную матрицу репарации используют для вставки последовательности в геном. В частных предпочтительных вариантах донорную матрицу репарации используют для репарации или модификации последовательности в геноме.
В различных вариантах реализации донорную матрицу репарации вводят в гемопоэтическую клетку, например Т-клетку, путем трансдукции клетки вектором адено-ассоциированного вируса (AAV), ретровируса, например, лентивируса, IDLV и т.д., вируса простого герпеса, аденовируса или вируса коровьей оспы, содержащим донорную матрицу репарации.
В частных вариантах реализации донорная матрица репарации содержит одно или более плеч гомологии, которые фланкируют сайт DSB.
Используемый в настоящей заявке термин «гомологичные плечи» относится к нуклеотидной последовательности в донорной матрице репарации, которая идентична или почти идентична последовательности ДНК, фланкирующей разрыв ДНК, введенный нуклеазой в сайте-мишени. В одном варианте реализации донорная матрица репарации содержит 5'-гомологичное плечо, которое содержит нуклеотидную последовательность, которая идентична или почти идентична последовательности ДНК 5' сайта разрыва ДНК. В одном варианте реализации донорная матрица репарации содержит 3'-гомологичное плечо, которое содержит нуклеотидную последовательность, которая идентична или почти идентична последовательности ДНК 3' сайта разрыва ДНК. В предпочтительном варианте реализации донорная матрица репарации содержит 5'-гомологичное плечо и 3'-гомологичное плечо. Донорная матрица репарации может содержать гомологию с геномной последовательностью, непосредственно примыкающей к сайту DSB, или гомологию с геномной последовательностью в пределах любого количества пар оснований из сайта DSB. В одном варианте реализации донорная матрица репарации содержит нуклеотидную последовательность, которая гомологична геномной последовательности длиной приблизительно 5 п.н., приблизительно 10 п.н., приблизительно 25 п.н., приблизительно 50 п.н., приблизительно 100 п.н., приблизительно 250 п.н., приблизительно 500 п.н., приблизительно 1000 п.н., приблизительно 2500 п.н., приблизительно 5000 п.н., приблизительно 10000 п.н.или более, включая любую промежуточную длину гомологичной последовательности.
Иллюстративные примеры подходящих длин гомологичных плеч, рассматриваемых в частных вариантах реализации могут быть выбраны независимо и включают, среди прочего: гомологичные плечи длиной приблизительно 100 п.н., приблизительно 200 п.н., приблизительно 300 п.н., приблизительно 400 п.н., приблизительно 500 п.н., приблизительно 600 п.н., приблизительно 700 п.н., приблизительно 800 п.н., приблизительно 900 п.н., приблизительно 1000 п.н., приблизительно 1100 п.н., приблизительно 1200 п.н., приблизительно 1300 п.н., приблизительно 1400 п.н., приблизительно 1500 п.н., приблизительно 1600 п.н., приблизительно 1700 п.н., приблизительно 1800 п.н., приблизительно 1900 приблизительно 2000 п.н., приблизительно 2100 п.н., приблизительно 2200 п.н., приблизительно 2300 п.н., приблизительно 2400 п.н., приблизительно 2500 п.н., приблизительно 2600 п.н., приблизительно 2700 п.н., приблизительно 2800 п.н., приблизительно 2900 п.н. или приблизительно 3000 п.н., или больше, включая все промежуточные длины гомологичных плеч.
Дополнительные иллюстративные примеры подходящей длины гомологичного плеча включают, среди прочего: от приблизительно 100 до приблизительно 3000 п.н., от приблизительно 200 до приблизительно 3000 п.н., от приблизительно 300 до приблизительно 3000 п.н., от приблизительно 400 до приблизительно 3000 п.н., приблизительно 500 от приблизительно 3000 до приблизительно 2500 п.н., от приблизительно 500 до приблизительно 2500 п.н., от приблизительно 500 до приблизительно 2000 п.н., от приблизительно 750 до приблизительно 2000 п.н., от приблизительно 750 до приблизительно 1500 п.н. или от приблизительно 1000 до приблизительно 1500 п.н., включая все промежуточные длины гомологичных плеч.
В частном варианте реализации длины 5'- и 3'-гомологичных плечи независимо выбраны от приблизительно 500 до приблизительно 1500 п.н. В одном варианте реализации 5'-гомологичное плечо имеет длину приблизительно 1500 п.н., а 3'-гомологичное плечо имеет длину приблизительно 1000 п.н. В одном варианте реализации 5' гомологичное плечо имеет длину от 200 до 600 п.н., а гомологичное плечо имеет длину от 200 до 600 п.н. В одном варианте реализации 5'-гомологичное плечо имеет длину приблизительно 200 п.н., а 3'-гомологичное плечо имеет длину приблизительно 200 п.н. В одном варианте 5'гомологичное плечо имеет длину приблизительно 300 п.н., а 3'-гомологичное плечо имеет длину приблизительно 300 п.н. В одном варианте реализации 5'-гомологичное плечо имеет длину приблизительно 400 п.н., а 3'-гомологичное плечо имеет длину приблизительно 400 п.н. В одном варианте 5'гомологичное плечо имеет длину приблизительно 500 п.н., а 3'-гомологичное плечо имеет длину приблизительно 500 п.н. В одном варианте реализации 5'-гомологичное плечо имеет длину приблизительно 600 п.н., а 3'-гомологичное плечо имеет длину приблизительно 600 п.н.
Донорная матрица репарации может, кроме того, содержать один или более полинуклеотидов, таких как промоторы и/или энхансеры, нетранслируемые области (UTR), последовательности Козака, сигналы полиаденилирования, дополнительные сайты ферментов рестрикции, множественные сайты клонирования, участки внутренней посадки рибосомы (IRES), сайты узнавания рекомбиназы. (например, сайты LoxP, FRT и Att), терминирующие кодоны, сигналы терминации транскрипции и полинуклеотиды, кодирующие саморасщепляемые полипептиды, метки эпитопа, рассматриваемые в других местах настоящей заявки.
В одном варианте реализации донорная матрица репарации содержит полинуклеотид, содержащий ген PD-1, или его часть, и предназначена для введения одной или нескольких мутаций в геномную последовательность PD-1 таким образом, чтобы экспрессировать продукт мутантного гена PD-1. В одном варианте реализации мутант PD-1 обладает сниженным связыванием лиганда и/или уменьшением внутриклеточной передачи сигналов.
В различных вариантах реализации донорная матрица репарации содержит 5'-гомологичное плечо, промотор РНК-полимеразы II, один или более полинуклеотидов, кодирующих энхансер иммунопотенции, демпфер иммуносупрессивного сигнала или рецептор сконструированного антигена и 3'-гомологичное плечо.
В различных вариантах реализации сайт-мишень модифицирован донорной матрицей репараций, содержащей 5'-гомологичное плечо, один или более полинуклеотидов, кодирующих саморасщепляемый вирусный пептид, например, T2A, энхансер иммунопотенции, демпфер иммуносупрессивного сигнала или рецептор сконструированного антигена, необязательно поли(А)-сигнал или саморасщепляемый пептид, и 3'-гомологичное плечо, причем экспрессия одного или нескольких полинуклеотидов регулируется эндогенным промотором PD-1.
1. Энхансеры иммунопотенции
В частных вариантах реализации рассматриваемые в настоящей заявке иммунные эффекторные клетки с отредактированным геномом делаются более активными и/или устойчивыми к иммуносупрессивным факторам путем введения DSB в ген PD-1 в присутствии донорной матрицы репарации, кодирующей энхансер иммунопотенции. Используемый в настоящей заявке термин «энхансер иммунопотенции» относится к не встречающимся в природе молекулам, которые стимулируют и/или потенцируют активацию и/или функцию Т-клеток, иммунопотенцирующим факторам и не встречающимся в природе полипептидам, которые преобразуют иммуносупрессивные сигналы из микроокружения опухоли в иммуностимулирующий сигнал в Т-клетке или других иммунных клетках.
В частных вариантах реализации энхансер иммунопотенции выбран из группы, состоящей из молекулы биспецифического Т-клеточного активатора (BiTE); иммуностимулирующего фактора, включая, среди прочего, цитокины, хемокины, цитотоксины и/или рецепторы цитокинов; и флип-рецептор.
В некоторых вариантах реализации энхансер иммунопотенции, иммунопотенцирующий фактор или флип-рецептор представляют собой слитые полипептиды, содержащие домен дестабилизации белка.
a. Молекулы биспецифического Т-клеточного активатора (BiTE)
В частных вариантах реализации рассматриваемые в настоящей заявке иммунные эффекторные клетки с отредактированным геномом становятся более эффективными при помощи введения DSB в ген PD-1 в присутствии донорной матрицы репарации, кодирующей молекулы биспецифического Т-клеточного активатора (BiTE). Молекулы BiTE представляют собой молекулы из двух частей, содержащие первый связывающий домен, который связывает антиген-мишень, линкер или спейсер, рассматриваемый в другом месте настоящей заявки, и второй связывающий домен, который связывает стимулирующую или костимулирующую молекулу на иммунной эффекторной клетке. Первый и второй связывающий домены могут быть независимо выбраны из лигандов, рецепторов, антител или их антигенсвязывающих фрагментов, лектинов и углеводов.
В частных вариантах реализации первый и второй связывающие домены представляют собой антиген-связывающий домены.
В частных вариантах реализации первый и второй связывающие домены представляют собой антитела или их антигенсвязывающие фрагменты. В одном варианте реализации первый и второй связывающие домены представляют собой одноцепочечные вариабельные фрагменты (scFv).
Иллюстративные примеры антигенов-мишеней, которые могут распознаваться и связываться первым связывающим доменом в частных вариантах реализации, включают, среди прочего: рецептор альфа-фолата, 5T4, интегрин αvβ6, BCMA, B7-H3, B7-H6, CAIX, CD16, CD19, CD20, CD22, CD30, CD33, CD44, CD44v6, CD44v7/8, CD70, CD79a, CD79b, CD123, CD138, CD171, CEA, CSPG4, EGFR, семейство EGFR включая ErbB2 (HER2), EGFRvIII, EGP2, EGP40, EPCAM, EphA2, EpCAM, FAP, фетальный AchR, FRα, GD2, GD3, Glypican-3 (GPC3), HLA-A1+MAGE1, HLA-A2+MAGE1, HLA-A3+MAGE1, HLA-A1+NY-ESO-1, HLA-A2+NY-ESO-1, HLA-A3+NY-ESO-1, IL-11Rα, IL-13Rα2, Лямбда, Lewis-Y, Каппа, Мезотелин, Muc1, Muc16, NCAM, лиганды NKG2D, NY-ESO-1, PRAME, PSCA, PSMA, ROR1, SSX, сурвивин, TAG72, TEMs, VEGFR2 и WT-1.
Другие иллюстративные варианты реализации антигенов-мишеней включают комплексы MHC-пептид, необязательно в которых пептид процессирован из альфа-фолатного рецептора, 5T4, интегрина αvβ6, BCMA, B7-H3, B7-H6, CAIX, CD16, CD19, CD20, CD22, CD30, CD33, CD44, CD44v6, CD44v7/8, CD70, CD79a, CD79b, CD123, CD138, CD171, CEA, CSPG4, EGFR, семейства EGFR включая ErbB2 (HER2), EGFRvIII, EGP2, EGP40, EPCAM, EphA2, EpCAM, FAP, фетального AchR, FRα, GD2, GD3, Glypican-3 (GPC3), MAGE1, NY-ESO-1, IL-11Rα, IL-13Rα2, Лямбда, Lewis-Y, Каппа, Мезотелин, Muc1, Muc16, NCAM, лиганды NKG2D, PRAME, PSCA, PSMA, ROR1, SSX, сурвивин, TAG72, TEMs, VEGFR2 и WT-1.
Иллюстративные примеры стимулирующих или костимулирующих молекул на эффекторных клетках иммунитета, распознаваемых и связываемых вторым связывающим доменом в частных вариантах реализации, включают, среди прочего: CD3γ, CD3δ, CD3ε, CD3ζ, CD28, CD134, CD137 и CD278.
В частных вариантах реализации DSB в гене PD-1 индуцируют сконструированной нуклеазой, а донорную матрицу репарации, содержащую BiTE, вводят в клетку и встраивают в ген PD-1 путем гомологичной рекомбинации.
b. Иммунопотенцирующие факторы
В частных вариантах реализации рассматриваемые в настоящей заявке иммунные эффекторные клетки с отредактированным геномом становятся более активными за счет увеличения иммунопотенцирующих факторов либо в клетках с отредактированным геномом, либо в клетках в микроокружении опухоли. «Иммунопотенцирующие факторы» относятся к конкретным цитокинам, хемокинам, цитотоксинам и рецепторам цитокинов, которые усиливают иммунный ответ в иммунных эффекторных клетках. В одном варианте реализации Т-клетки сконструированы путем введения DSB в ген PD-1 в присутствии донорной матрицы репарации, кодирующей рецептор цитокина, хемокина, цитотоксина или цитокина.
В частных вариантах реализации донорная матрица репарации кодирует цитокин, выбранный из группы, состоящей из: IL-2, инсулина, IFN-γ, IL-7, IL-21, IL-10, IL-12, IL-15 и TNF-α.
В предпочтительном варианте реализации донорная матрица репарации кодирует цитокин, выбранный из группы, состоящей из IL-2, IL-7, IL-12, IL-15, IL-18 и IL-21, который будучи встроен в сайт-мишень в гене PD-1, функционально связывает цитокин с эндогенным промотором PD-1, тем самым помещая транскрипционный контроль цитокина под контроль эндогенного промотора PD-1.
В другом предпочтительном варианте реализации донорная матрица репарации кодирует IL-12, который, будучи встроен в сайт-мишень в гене PD-1, функционально связывает цитокин с эндогенным промотором PD-1, тем самым помещая транскрипционный контроль цитокина под контроль эндогенного промотора PD-1.
В частных вариантах реализации донорная матрица репарации кодирует хемокин, выбранный из группы, состоящей из: MIP-1α, MIP-1β, MCP-1, MCP-3 и RANTES.
В частных вариантах реализации донорная матрица репарации кодирует цитотоксин, выбранный из группы, состоящей из перфорина, гранзима A и гранзима B.
В частных вариантах реализации донорная матрица репарации кодирует рецептор цитокина, выбранный из группы, состоящей из: рецептора IL-2, рецептора IL-7, рецептора IL-12, рецептора IL-15 и рецептора IL-21.
В предпочтительном варианте реализации донорная матрица репарации кодирует рецептор цитокина, выбранный из группы, состоящей из рецептора IL-2, рецептора IL-7, рецептора IL-12, рецептора IL-15, рецептора IL-18 и рецептора IL-21, который, будучи встроен в сайт-мишень в гене PD-1 функционально связывает рецептор цитокинов с эндогенным промотором PD-1, тем самым помещая транскрипционный контроль рецептора цитокинов под контроль эндогенного PD- 1 промоутер.
В другом предпочтительном варианте реализации донорная матрица репарации кодирует рецептор IL-12, который, будучи встроен в сайт-мишень в гене PD-1, оперативно связывает рецептор цитокина с эндогенным промотором PD-1, тем самым помещая транскрипционный контроль рецептора цитокинов под контроль эндогенного под контролем эндогенного промотора PD-1.
c. Флип-рецепторы
В других вариантах реализации донорная матрица репарации кодирует флип-рецептор или его часть. Используемый в настоящей заявке термин «флип-рецептор» относится к не встречающемуся в природе полипептиду, который преобразует иммуносупрессивные сигналы из микроокружения опухоли в иммуностимулирующий сигнал в Т-клетке. В частных вариантах реализации «флип-рецептор PD-1» относится к полипептиду, который содержит экзодомен PD-1 или связывающий домен лиганда или его вариант, трансмембранный домен и эндодомен, который трансдуцирует иммуностимулирующий сигнал в Т-клетку. В частных вариантах реализации «флип-рецептор PD-1» относится к полипептиду, который содержит экзодомен PD-1 или связывающий домен лиганда или его вариант, трансмембранный домен PD-1 и эндодомен, который трансдуцирует иммуностимулирующий сигнал в Т-клетку. В частных вариантах реализации «флип-рецептор PD-1» относится к полипептиду, который содержит экзодомен PD-1 или связывающий домен лиганда или его вариант и трансмембранный домен и эндодомен из белка, который трансдуцирует иммуностимулирующий сигнал в Т-клетку. В некоторых вариантах реализации вариант экзодомена PD-1 обладает повышенной аффинностью связывания с PD-L1 и/или PD-L2.
В одном варианте реализации трансмембрана выделена из CD3δ, CD3ε, CD3γ, CD3ζ, CD4, CD5, CD8α, CD9, CD 16, CD22, CD27, CD28, CD33, CD37, CD45, CD64, CD80, CD86, CD 134, CD137, CD152, CD154, AMN и PD-1.
В одном варианте реализации трансмембрана выделена из CD4, CD8α, CD8β, CD27, CD28, CD134, CD137, полипептида CD3, рецептора IL-2, рецептора IL-7, рецептора IL-12, рецептора IL-15 или рецептора IL- 21.
В одном варианте реализации эндодомен выделен из рецептора IL-2, рецептора IL-7, рецептора IL-12, рецептора IL-15 или рецептора IL-21.
В одном варианте реализации донорная матрица репарации содержит флип-рецептор PD-1, содержит экзодомен или связывающий домен лиганда PD-1, трансмембранный домен и один или более внутриклеточных костимулирующих сигнальных доменов и/или первичных сигнальных доменов. Трансмембранные и эндодомены могут быть выделены из одного и того же белка или разных белков.
В одном варианте реализации донорная матрица репарации содержит флип-рецептор PD-1, содержит экзодомен или лиганд связывающий домен PD-1, трансмембранный домен PD-1 и один или более внутриклеточных костимуляторных сигнальных доменов и/или первичных сигнальных доменов.
2. Демпферы иммуносупрессивного сигнала
Одним из ограничений или проблем, которые являются слабым местом существующей адоптивной клеточной терапии, является гипореактивность иммунных эффекторных клеток вследствие истощения, опосредованного микроокружением опухоли. Истощенные Т-клетки имеют уникальную молекулярную сигнатуру, которая заметно отличается от наивных, эффекторных Т-клеток или Т-клеток памяти. Истощенные Т-клетки определяются как Т-клетки со снижением экспрессии цитокинов и эффекторной функции.
В частных вариантах реализации рассматриваемые в настоящей заявке иммунные эффекторные клетки с отредактированным геномом становятся более устойчивыми к истощению за счет снижения или демпфирования передачи сигналов иммунодепрессивными факторами. В одном варианте реализации Т-клетки сконструированы путем введения DSB в ген PD-1 в присутствии донорной матрицы репарации, кодирующей демпфер иммуносупрессивного сигнала.
Используемый в настоящей заявке термин «демпфер иммуносупрессивного сигнала» относится к не встречающемуся в природе полипептиду, который уменьшает передачу иммуносупрессивных сигналов из микроокружения опухоли в Т-клетку. В одном варианте реализации «демпфер иммуносупрессивного сигнала» представляет собой антитело или его антигенсвязывающий фрагмент, который связывает иммуносупрессивный фактор. В предпочтительных вариантах реализации «демпфер иммуносупрессивного сигнала» относится к полипептиду, который оказывает супрессивный, демпфирующий или доминирующий отрицательный эффект на конкретный иммуносупрессивный фактор или путь передачи сигнала, поскольку демпфер содержит экзодомен, который связывает иммуносупрессивный фактор и, необязательно, трансмембранный домен, и, необязательно, модифицированный эндодомен (например, внутриклеточный сигнальный домен).
В частных вариантах реализации экзодомен представляет собой внеклеточный связывающий домен, который распознает и связывает и иммуносупрессивный фактор.
В частных вариантах реализации такой модифицированный эндодомен мутирован для уменьшения или ингибирования иммуносупрессивных сигналов. Подходящие стратегии мутации включают, среди прочего, замену, добавление или делецию аминокислоты. Подходящие мутации дополнительно включают, среди прочего, укорочение эндодомена для удаления сигнальных доменов, мутацию эндодоменов для удаления остатков, важных для активности сигнального мотива, и мутацию эндодоменов для блокировки циклирования рецепторов. В частных вариантах реализации эндодомен, когда он присутствует, не трансдуцирует иммуносупрессивные сигналы или обладает существенно сниженной способностью трансдуцировать иммуносупрессивные сигналы.
Таким образом, в некоторых вариантах реализации демпфер иммуносупрессивного сигнала действует как поглотитель для одного или нескольких иммуносупрессивных факторов из микроокружения опухоли и ингибирует соответствующие пути передачи иммуносупрессивных сигналов в Т-клетке.
Один иммуносупрессивный сигнал опосредован катаболизмом триптофана. Катаболизм триптофана индоламиновой 2,3-диоксигеназой (IDO) в раковых клетках приводит к выработке кинуренинов, которые, как было показано, оказывают иммуносупрессивное действие на Т-клетки в микроокружении опухоли. См. например, Platten et al. (2012) Cancer Res. 72(21):5435-40.
В одном варианте реализации донорная матрица репарации содержит фермент с кинурениназной активностью.
Иллюстративные примеры ферментов, обладающих кинурениназной активностью, подходящих для применения в частных вариантах реализации, включают, среди прочего, L-кинуренингидролазу.
В одном варианте реализации донорная матрица репарации содержит один или более полинуклеотидов, кодирующих демпфер иммуносупрессивного сигнала, который уменьшает или блокирует передачу иммуносупрессивных сигналов, опосредованную иммуносупрессивным фактором.
Иллюстративные примеры иммуносупрессивных факторов, на которые нацелены демпферы иммуносупрессивного сигнала, рассматриваемые в частных вариантах реализации включают, среди прочего: лиганд запрограммированной смерти 1 (PD-L1), лиганд запрограммированной смерти 2 (PD-L2), трансформирующий фактор роста β (TGFβ), макрофагальный колониестимулирующий фактор 1 (M-CSF1), родственный фактору некроза опухоли лиганд, индуцирующий апоптоз (TRAIL), рецептор-связывающий раковый антиген, экспрессированный на лиганде клеток SiSo (RCAS1), лиганд Fas (FasL), CD47, интерлейкин-4 (IL-4), интерлейкин-6 (IL-6), интерлейкин-8 (IL-8), интерлейкин-10 (IL-10) и интерлейкин-13 (IL-13).
В различных вариантах реализации демпфер иммуносупрессивного сигнала содержит антитело или его антигенсвязывающий фрагмент, который связывает иммуносупрессивный фактор.
В различных вариантах реализации демпфер иммуносупрессивного сигнала содержит экзодомен, который связывает иммуносупрессивный фактор.
В частных вариантах реализации демпфер иммуносупрессивного сигнала содержит экзодомен, который связывает иммуносупрессивный фактор и трансмембранный домен.
В другом варианте реализации демпфер иммуносупрессивного сигнала содержит экзодомен, который связывает иммуносупрессивный фактор, трансмембранный домен и модифицированный эндодомен, который не трансдуцирует или который обладает существенно сниженной способностью трансдуцировать иммуносупрессивные сигналы.
Используемый в настоящей заявке термин «экзодомен» относится к антиген-связывающему домену. В одном варианте реализации экзодомен представляет собой внеклеточный лиганд-связывающий домен иммуносупрессивного рецептора, который трансдуцирует иммуносупрессивные сигналы из микроокружения опухоли в Т-клетку. В частных вариантах реализации «экзодомен» относится к внеклеточному лиганд- связывающему домену рецептора, который содержит иммунорецепторный тирозиновый ингибирующий мотив (ITIM) и/или иммунорецепторный мотив переключения тирозина (ITSM).
Иллюстративные примеры экзодоменов, подходящих для применения в частных вариантах реализации демпферов иммуносупрессивного сигнала, включают, среди прочего, антитела или их антигенсвязывающие фрагменты или внеклеточные лиганд-связывающие домены, выделенные из следующих полипептидов: белок запрограммированной гибели клеток 1 (PD-1), белок гена активации лимфоцитов 3 (LAG-3), домен Т-клеточного иммуноглобулина и белок 3 муцинового домена (TIM3), антиген 4 цитотоксических Т лимфоцитов 4 (CTLA-4), B- и T-лимфоцитарный аттенюатор (BTLA), иммуноглобулин Т-клеток и иммунорецепторный домен ингибирующий мотива на основе тирозина (TIGIT), рецептор II трансформирующего фактора роста β (TGFβRII), рецептор макрофагального колониестимулирующего фактора 1 (CSF1R), рецептор интерлейкина 4 (IL4R), рецептор интерлейкина 6 (IL6R), рецептор хемокина (мотив C-X-C) 1 (CXCR1), рецептор 2 хемокина (мотив C-X-C) (CXCR2), субъединица альфа-рецептора интерлейкина 10 (IL10R), субъединица рецептора интерлейкина 13 альфа-2 (IL13Rα2), рецептор к апоптоз-индуцирующему лиганду семейства TNF (TRAILR1), рецептор-связывающий раковый антиген, экспрессируемый на клетках SiSo (RCAS1R), и рецептор гибели на поверхности клетки Fas (FAS).
В одном варианте реализации экзодомен содержит внеклеточный лиганд-связывающий домен рецептора, выбранный из группы, состоящей из: PD-1, LAG-3, TIM3, CTLA-4, IL10R, TIGIT, CSF1R и TGFβRII.
В частных вариантах реализации может быть использован ряд трансмембранных доменов. Иллюстративные примеры трансмембранных доменов, подходящих для применения в частных вариантах реализации демпферов иммуносупрессивного сигнала, рассматриваемых в частных вариантах реализации включают, среди прочего, трансмембранные домены следующих белков: альфа- или бета-цепь рецептора Т-клеток, CDδ, CD3ε, CDγ, CD3ζ, CD4, CD5, CD8α, CD9, CD 16, CD22, CD27, CD28, CD33, CD37, CD45, CD64, CD80, CD86, CD 134, CD137, CD152, CD154 и PD-1.
В частных вариантах реализации рассматриваемая в настоящей заявке адоптивная клеточная терапия включает в себя демпфер иммуносупрессивного сигнала, который ингибирует или блокирует трансдукцию иммуносупрессивных сигналов TGFβ из микроокружения опухоли через TGFβRII. В одном варианте реализации демпфер иммуносупрессивного сигнала содержит экзодомен, который содержит связывание внеклеточного лиганда TGFβRII, трансмембранный домен TGFβRII и усеченный нефункциональный эндодомен TGFβRII. В другом варианте реализации демпфер иммуносупрессивного сигнала содержит экзодомен, который содержит связывание внеклеточного лиганда TGFβRII, трансмембранный домен TGFβRII и не имеет эндодомена.
3. Сконструированные антигенные рецепторы
В частных вариантах реализации рассматриваемые в настоящей заявке иммунные эффекторные клетки с отредактированным геномом содержат сконструированный антигенный рецептор. В одном варианте реализации Т-клетки сконструированы сконструированным способом путем введения DSB в один или более генов PD-1 в присутствии донорной матрицы репарации, кодирующей сконструированный антигенный рецептор.
В частных вариантах реализации сконструированный антигенный рецептор представляет собой сконструированный рецептор Т-клеток (TCR), химерный антигенный рецептор (CAR), рецептор Daric или его компоненты или химерный рецептор цитокинов.
a. Сконструированные TCR
В частных вариантах реализации рассматриваемые в настоящей заявке с отредактированным геномом иммунные эффекторные клетки содержат сконструированный TCR. В одном варианте реализации Т-клетки сконструированы путем введения DSB в один или более генов PD-1 в присутствии донорной матрицы репарации, кодирующей сконструированный TCR. В частном варианте реализации сконструированный TCR вставляют в DSB в одном гене PD-1. В другом варианте альфа-цепь сконструированного TCR вставляют в DSB в одном гене PD-1, а бета-цепь сконструированного TCR вставляют в DSB в другом гене PD-1.
В одном варианте реализации сконструированные T-клетки, рассматриваемые в настоящей заявке, содержат сконструированный TCR, который не встроен в ген PD-1, и один или более из: демпфера иммуносупрессивного сигнала, флип-рецептора, альфа- и/или бета-цепи сконструированного Т-клеточного рецептора (TCR), химерного антигенного рецептора (CAR), рецептора Daric или его компонентов или химерного цитокинового рецептора вставляют в DSB в одном или нескольких генах PD-1.
Встречающиеся в природе рецепторы Т-клеток содержат две субъединицы: субъединицу альфа-цепи и бета-цепи, каждая из которых является уникальным белком, продуцируемым в результате рекомбинации в геноме каждой Т-клетки. Библиотеки TCR могут быть подвергнуты скринингу на их селективность к конкретным антигенам-мишеням. Таким образом, природные TCR, которые обладают высокой авидностью и реактивностью по отношению к антигенам-мишеням, могут быть отобраны, клонированы и впоследствии введены в популяцию Т-клеток, используемых для адоптивной иммунотерапии.
В одном варианте реализации Т-клетки модифицируют путем введения донорной матрицы репарации, содержащей полинуклеотид, кодирующий субъединицу TCR в DSB в одном или нескольких генах PD-1, причем указанная субъединица TCR обладает способностью образовывать TCR, которые придают специфичность Т-клеткам против опухолевых клеток, экспрессирующих антиген-мишень. В частных вариантах реализации эти субъединицы имеют одну или более аминокислотных замен, делеций, инсерций или модификаций по сравнению с встречающейся в природе субъединицей, при условии, что субъединицы сохраняют способность образовывать TCR и наделяют трансфицированные T-клетки способностью хоминга в направлении клеток-мишеней, и участвуют в иммунологически релевантной передаче сигналов цитокинов. Эти сконструированные TCR предпочтительно также связывают клетки-мишени, демонстрируя соответствующий опухоль-ассоциированный пептид с высокой авидностью, и необязательно обеспечивают эффективное уничтожение клеток-мишеней, презентирующих соответствующий пептид in vivo.
Нуклеиновые кислоты, кодирующие сконструированный TCR, предпочтительно выделяют из их естественного контекста во (встречающейся в природе) хромосоме Т-клетки и могут быть встроены в подходящие векторы, как описано в другом месте настоящей заявки. Как нуклеиновые кислоты, так и векторы, которые их содержат, могут быть перенесены в клетку, предпочтительно в Т-клетку в частных вариантах реализации. Такие модифицированные Т-клетки затем способны экспрессировать одну или более цепей TCR, кодируемых трансдуцированной нуклеиновой кислотой или нуклеиновыми кислотами. В предпочтительных вариантах реализации сконструированный TCR представляет собой экзогенный TCR, потому что он введен в T-клетки, которые обычно не экспрессируют данный конкретный TCR. Существенным аспектом этих сконструированных TCR является то, что он обладает высокой авидностью к опухолевому антигену, представленному основным комплексом гистосовместимости (MHC) или аналогичным иммунологическим компонентом. В отличие от сконструированного TCR, CAR сконструированы так, чтобы связывать целевые антигены MHC-независимым способом.
TCR может быть экспрессирован дополнительными полипептидами, присоединенными к аминоконцевой или карбоксил-концевой части альфа-цепи или бета-цепи по настоящему изобретению TCR, если только присоединенный дополнительный полипептид не влияет на способность альфа-цепи или бета-цепи сформировать функциональный Т-клеточный рецептор и MHC-зависимое распознавание антигена.
Антигены, которые распознаются сконструированным TCR, рассматриваемыми в частных вариантах реализации, включают, среди прочего, антигены рака, включая антигены как при гематологическом раке, так и в солидных опухолях. Иллюстративные антигены включают, среди прочего, альфа-фолатный рецептор, альфа-фолатный рецептор, 5T4, интегрин αvβ6, BCMA, B7-H3, B7-H6, CAIX, CD19, CD20, CD22, CD30, CD33, CD44, CD44v6, CD44v7/8, CD70, CD79a, CD79b, CD123, CD138, CD171, CEA, CSPG4, EGFR, семейство EGFR включая ErbB2 (HER2), EGFRvIII, EGP2, EGP40, EPCAM, EphA2, EpCAM, FAP, фетальный AchR, FRα, GD2, GD3, глипикан-3 (GPC3), HLA-A1+MAGE1, HLA-A2+MAGE1, HLA-A3+MAGE1, HLA-A1+NY-ESO-1, HLA-A2+NY-ESO-1, HLA-A3+NY-ESO-1, IL-11Rα, IL-13Rα2, лямбда, Lewis-Y, Каппа, мезотелин, Muc1, Muc16, NCAM, лиганды NKG2D, NY-ESO-1, PRAME, PSCA, PSMA, ROR1, SSX, сурвивин, TAG72, TEMs, VEGFR2 и WT-1.
В одном варианте реализации донорная матрица репарации содержит полинуклеотид, кодирующий промотор РНК-полимеразы II или первый саморасщепляемый вирусный пептид, и полинуклеотид, кодирующий альфа-цепь и/или бета-цепь сконструированного TCR, встроенного в один модифицированный и/или нефункциональный ген PD-1.
В одном варианте реализации донорная матрица репарации содержит полинуклеотид, кодирующий промотор РНК-полимеразы II или первый саморасщепляемый вирусный пептид, и полинуклеотид, кодирующий альфа-цепь и бета-цепь сконструированного TCR, встроенные в один модифицированный и/или нефункциональный ген PD-1.
В частном варианте реализации донорная матрица репарации содержит в направлении от 5' до 3', полинуклеотид, кодирующий первый саморасщепляемый вирусный пептид, полинуклеотид, кодирующий альфа-цепь сконструированного TCR, полинуклеотид, кодирующий второй саморасщепляемый вирусный пептид, и полинуклеотид, кодирующий бета-цепь сконструированного TCR, встроенные в один модифицированный и/или нефункциональный ген PD-1. В таком случае другой ген PD-1 может быть функциональным или может иметь пониженную функцию или быть лишенным функциональности посредством DSB и репарации методом NHEJ. В одном варианте реализации другой ген PD-1 был модифицирован сконструированной нуклеазой, рассматриваемой в настоящей заявке, и может иметь пониженную функцию или быть лишенным функциональности.
В определенном варианте реализации оба гена PD-1 модифицированы и имеют пониженную функцию или являются нефункциональными: первый модифицированный ген PD-1 содержит нуклеиновую кислоту, включающую полинуклеотид, кодирующий первый саморасщепляемый вирусный пептид, и полинуклеотид, кодирующий альфа-цепь сконструированный TCR, а второй модифицированный ген PD-1 содержит полинуклеотид, кодирующий второй саморасщепляемый вирусный пептид, и полинуклеотид, кодирующий бета-цепь сконструированного TCR.
b. Химерные антигенные рецепторы (CAR)
В частных вариантах реализации сконструированные иммунные эффекторные клетки, рассматриваемые в настоящей заявке, содержат один или более химерных антигенных рецепторов (CAR). В одном варианте реализации Т-клетки сконструированы путем введения DSB в один или более генов PD-1 в присутствии донорной матрицы репарации, кодирующей CAR. В частном варианте реализации CAR вставляют в DSB в одном гене PD-1.
В одном варианте реализации сконструированные T-клетки, рассматриваемые в настоящей заявке, содержат CAR, который не встроен в ген PD-1 и одно или более из: демпфера иммуносупрессивного сигнала, флип-рецептора, альфа- и/или бета-цепи сконструированного Т-клеточного рецептора (TCR), химерного цитокинового рецептора (CAR), рецептора Daric или их компонентов или химерный антигенный рецептор встроен в DSB в одном или нескольких генах PD-1.
В различных вариантах реализации Т-клетки с отредактированным геномом экспрессируют CAR, которые перенаправляют цитотоксичность на опухолевые клетки. CAR представляют собой молекулы, которые сочетают основанную на антителах специфичность к антигену-мишени (например, опухолевому антигену) с внутриклеточным доменом, активирующим рецептор Т-клеток, чтобы генерировать химерный белок, который проявляет специфическую противоопухолевую клеточную иммунную активность. Используемый в настоящей заявке термин «химерный» указывает, что белок состоит из частей разных белков или ДНК различного происхождения.
В различных вариантах реализации CAR содержит внеклеточный домен, который связывается со специфическим антигеном-мишенью (также называемым связывающим доменом или антигенспецифическим связывающим доменом), трансмембранным доменом и внутриклеточным сигнальным доменом. Основными характеристиками CAR являются их способность перенаправлять специфичность иммунных эффекторных клеток, тем самым вызывая пролиферацию, выработку цитокинов, фагоцитоз или выработку молекул, которые могут опосредовать клеточную гибель экспрессирующей антиген клетки-мишени независимым от главного комплекса гистосовместимости (MHC) способом, используя способности к специфическому нацеливанию на клетку моноклональных антител, растворимых лигандов или клетко-специфических корецепторов.
В частных вариантах реализации CAR содержат внеклеточный связывающий домен, который специфически связывается с полипептидом-мишенью, например, антигеном-мишенью, экспрессируемым в опухолевой клетке. Используемые в настоящей заявке термины «связывающий домен», «внеклеточный домен», «внеклеточный связывающий домен», «антиген-связывающий домен», «антигенспецифический связывающий домен» и «внеклеточный антигенспецифический связывающий домен» используются взаимозаменяемо и обозначают химерный рецептор, например, CAR или Daric, со способностью специфически связываться с рассматриваемым антигеном-мишенью. Связывающий домен может содержать любой белок, полипептид, олигопептид или пептид, который обладает способностью специфически распознавать и связываться с биологической молекулой (например, рецептором клеточной поверхности или опухолевым белком, липидом, полисахаридом или другой целевой молекулой клеточной поверхности, или его компонентом). Связывающий домен включает любой встречающийся в природе синтетический, полусинтетический или рекомбинантно полученный связывающий партнер для рассматриваемой биологической молекулы.
В частных вариантах реализации внеклеточный связывающий домен содержит антитело или его антигенсвязывающий фрагмент.
Термин «антитело» относится к связывающему агенту, который представляет собой полипептид, содержащий по меньшей мере вариабельную область легкой или тяжелой цепи иммуноглобулина, которая специфически распознает и связывает эпитоп антигена-мишени, такого как пептид, липид, полисахарид или нуклеиновая кислота, содержащая антигенный детерминант, например, распознаваемые иммунной клеткой. Антитела включают антигенсвязывающие фрагменты, например, Camel Ig (антитело верблюдовых или его фрагмент VHH), Ig NAR, фрагменты Fab, фрагменты Fab', фрагменты F(ab)' 2, фрагменты F(ab)'3, Fv, одноцепочечный вариабельный фрагмент антитела Fv («scFv»), бис-scFv, (scFv) 2, миниантитело, диатело, триатело, тетратело, стабилизированный дисульфидом белок Fv («dsFv») и однодоменное антитело (sdAb, Nanobody) или другие соответствующие фрагменты антител. Термин также включает сконструированные формы, такие как химерные антитела (например, гуманизированные мышиные антитела), гетероконъюгатные антитела (такие как биспецифичные антитела) и их антигенсвязывающие фрагменты. См. также, Pierce Catalog and Handbook, 1994-1995 (Pierce Chemical Co., Rockford, IL); Kuby, J., Immunology, 3rd Ed., W. H. Freeman & Co., New York, 1997.
В одном предпочтительном варианте реализации связывающий домен представляет собой scFv.
В другом предпочтительном варианте реализации связывающий домен представляет собой антитело верблюдовых.
В частных вариантах реализации CAR содержит внеклеточный домен, который связывает антиген, выбранный из группы, состоящей из: рецептора альфа-фолата, 5T4, интегрина αvβ6, BCMA, B7-H3, B7-H6, CAIX, CD16, CD19, CD20, CD22, CD30, CD33, CD44, CD44v6, CD44v7/8, CD70, CD79a, CD79b, CD123, CD138, CD171, CEA, CSPG4, EGFR, семейства EGFR включая ErbB2 (HER2), EGFRvIII, EGP2, EGP40, EPCAM, EphA2, EpCAM, FAP, фетального AchR, FRα, GD2, GD3, глипикана-3 (GPC3), HLA-A1+MAGE1, HLA-A2+MAGE1, HLA-A3+MAGE1, HLA-A1+NY-ESO-1, HLA-A2+NY-ESO-1, HLA-A3+NY-ESO-1, IL-11Rα, IL-13Rα2, Лямбда, Lewis-Y, Каппа, мезотелина, Muc1, Muc16, NCAM, лигандов NKG2D, NY-ESO-1, PRAME, PSCA, PSMA, ROR1, SSX, сурвивина, TAG72, TEMs, VEGFR2 и WT-1.
В частных вариантах реализации CAR содержат внеклеточный связывающий домен, например антитело или его антигенсвязывающий фрагмент, который связывает антиген, причем указанный антиген представляет собой комплекс MHC-пептид, такой как комплекс MHC класса I-пептид или комплекс MHC класса II -пептид.
В некоторых случаях CAR содержат остатки линкера между различными доменами. «Связывающая последовательность вариабельных областей» представляет собой аминокислотную последовательность, которая связывает вариабельную область тяжелой цепи с вариабельной областью легкой цепи и обеспечивает функцию спейсера, совместимую с взаимодействием двух связывающих доменов, так что полученный полипептид сохраняет специфическую аффинность связывания с той же молекулой-мишенью, что и антитело, которое содержит те же вариабельные области легкой и тяжелой цепи. В частных вариантах реализации CAR содержат один, два, три, четыре или пять или более линкеров. В частных вариантах реализации длина линкера составляет от приблизительно 1 до приблизительно 25 аминокислот, от приблизительно 5 до приблизительно 20 аминокислот или от приблизительно 10 до приблизительно 20 аминокислот или любую промежуточную длину аминокислот.В некоторых вариантах реализации длина линкера составляет 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22 23, 24, 25 или более аминокислот.
В частных вариантах реализации за связывающим доменом CAR следует один или более «спейсерных доменов», т.е. областей, которые перемещает антиген связывающий домен прочь от поверхности эффекторных клеток, чтобы обеспечить надлежащий межклеточный контакт, связывание антигена и активацию (Patel et al., Gene Therapy, 1999; 6: 412-419). Спейсерный домен может быть получен из природного, синтетического, полусинтетического или рекомбинантного источника. В некоторых случаях спейсерный домен представляет собой часть иммуноглобулина, включая, среди прочего, одну или более константных областей тяжелой цепи, например, CH2 и CH3. Спейсерный домен может включать аминокислотную последовательность встречающейся в природе шарнирной области иммуноглобулина или измененную шарнирную область иммуноглобулина.
В одном варианте спейсерный домен содержит CH2 и CH3 иммуноглобулина IgG1, IgG4, или IgD.
В одном варианте реализации связывающий домен CAR связан с одним или несколькими «шарнирными доменами», которые играют свою роль в помещении антиген-связывающего домена вдали от поверхности эффекторных клеток, чтобы обеспечить надлежащий межклеточный контакт, связывание антигена и активацию. CAR обычно содержит один или более шарнирных доменов между связывающим доменом и трансмембранным доменом (TM). Шарнирный домен может быть получен из природного, синтетического, полусинтетического или рекомбинантного источника. Шарнирный домен может включать аминокислотную последовательность встречающейся в природе шарнирной области иммуноглобулина или измененную шарнирную область иммуноглобулина.
Иллюстративные шарнирные домены, подходящие для использования в CAR, описанных в настоящей заявке, включают шарнирную область, полученную из внеклеточных областей мембранных белков типа 1, таких как CD8α и CD4, которые могут быть шарнирными областями дикого типа из этих молекул или могут быть изменены. В другом варианте шарнирный домен содержит шарнирную область CD8α.
В одном варианте реализации шарнир представляет собой шарнир PD-1 или шарнир CD152.
«Трансмембранный домен» представляет собой часть CAR, которая объединяет внеклеточную связывающую часть и внутриклеточный сигнальный домен и прикрепляет CAR к плазматической мембране иммунной эффекторной клетки. Домен ТМ может быть получен из природного, синтетического, полусинтетического или рекомбинантного источника.
Иллюстративные домены ТМ могут быть получены из (то есть содержат по меньшей мере трансмембранную область (-и)) альфа- или бета-цепи рецептора Т-клеток, CD3δ, CD3ε, CD3γ, CD3ζ, CD4, CD5, CD8α, CD9, CD 16, CD22, CD27, CD28, CD33, CD37, CD45, CD64, CD80, CD86, CD 134, CD137, CD152, CD154, AMN θ PD-1.
В одном варианте реализации CAR содержит домен TM, полученный из CD8α. В другом варианте реализации рассматриваемый в настоящей заявке CAR содержит домен TM, полученный из CD8α, и короткий олиго- или полипептидный линкер, предпочтительно длиной 1, 2, 3, 4, 5, 6, 7, 8, 9 или 10 аминокислот, который связывает домен TM и домен внутриклеточной сигнализации CAR. Особенно подходящим линкером является глицин-сериновый линкер.
В частных вариантах реализации CAR содержит внутриклеточный сигнальный домен. Термин «внутриклеточный сигнальный домен» относится к части CAR, которая участвует в передаче сообщения об эффективном связывании CAR с антигеном-мишенью во внутреннюю часть иммунной эффекторной клетки, чтобы вызвать функцию эффекторных клеток, например, активацию, выработку цитокинов, пролиферацию и цитотоксическую активность, включая высвобождение цитотоксических факторов в CAR-связанную клетку-мишень или другие клеточные ответы, вызванные связыванием антигена с внеклеточным доменом CAR.
Термин «эффекторная функция» относится к специализированной функции клетки. Например, эффекторной функцией Т-клетки может быть цитолитическая активность или помощь или активность, включая секрецию цитокина. Таким образом, термин «внутриклеточный сигнальный домен» относится к части белка, которая трансдуцирует сигнал эффекторной функции и которая направляет клетку на выполнение специализированной функции. Хотя обычно можно использовать весь внутриклеточный сигнальный домен, во многих случаях нет необходимости использовать весь домен. В той степени, в которой используется усеченная часть внутриклеточного сигнального домена, такая усеченная часть может использоваться вместо всего домена, при условии, что она трансдуцирует сигнал эффекторной функции. Термин «внутриклеточный сигнальный домен» включает в себя любую усеченную часть внутриклеточного сигнального домена, достаточную для трансформации сигнала эффекторной функции.
Известно, что сигналов, генерируемых только через TCR, недостаточно для полной активации Т-клетки и что также необходим вторичный или костимулирующий сигнал. Таким образом, можно сказать, что активация Т-клеток опосредуется двумя различными классами внутриклеточных сигнальных доменов: первичными сигнальными доменами, которые инициируют антиген-зависимую первичную активацию через TCR (например, комплекс TCR/CD3), и костимулирующими сигнальными доменами, которые действуют антиген-независимым образом обеспечивая вторичный или костимулирующий сигнал. В предпочтительных вариантах реализации CAR содержит внутриклеточный сигнальный домен, который содержит один или более «костимулирующих сигнальных доменов» и «первичный сигнальный домен».
Первичные сигнальные домены регулируют первичную активацию комплекса TCR либо стимулирующим, либо ингибирующим образом. Первичные сигнальные домены, которые действуют стимулирующим образом, могут содержать сигнальные мотивы, известные как иммунорецепторные мотивы активации на основе тирозина или ITAM.
Иллюстративные примеры ITAM, содержащих первичные сигнальные домены, подходящие для использования в CAR, рассматриваемых в частных вариантах реализации включают в себя домены, полученные из FcRγ, FcRβ, CD3γ, CD3δ, CD3ε, CD3ζ, CD22, CD79a, CD79b и CD66d. В частных предпочтительных вариантах реализации CAR содержит первичный сигнальный домен CD3ζ и один или более костимуляторных сигнальных доменов. Внутриклеточные первичные сигнальные и костимулирующие сигнальные домены могут быть связаны в любом порядке в тандеме с карбоксильным концом трансмембранного домена.
В частных вариантах реализации CAR содержит один или более костимуляторных сигнальных доменов для усиления эффективности и экспансии Т-клеток, экспрессирующих рецепторы CAR. Используемый в настоящей заявке термин «костимуляторный сигнальный домен» или «костимуляторный домен» относится к внутриклеточному сигнальному домену костимулирующей молекулы.
Иллюстративные примеры таких костимулирующих молекул, подходящих для использования в CAR, рассматриваемых в частных вариантах реализации включают TLR1, TLR2, TLR3, TLR4, TLR5, TLR6, TLR7, TLR8, TLR9, TLR10, CARD11, CD2, CD7, CD27, CD28, CD30, CD40, CD54 (ICAM), CD83, CD134 (OX40), CD137 (4-1BB), CD278 (ICOS), DAP10, LAT, NKD2C, SLP76, TRIM и ZAP70. В одном варианте реализации CAR содержит один или более костимуляторных сигнальных доменов, выбранных из группы, состоящей из CD28, CD137 и CD134, и первичного сигнального домена CD3ζ.
В различных вариантах реализации CAR содержит: внеклеточный домен, который связывает антиген, выбранный из группы, состоящей из: BCMA, CD19, CSPG4, PSCA, ROR1 и TAG72; трансмембранный домен, выделенный из полипептида, выбранного из группы, состоящей из: CD4, CD8α, CD154 и PD-1; один или более внутриклеточных костимулирующих сигнальных доменов, выделенных из полипептида, выбранного из группы, состоящей из: CD28, CD134 и CD137; и сигнальный домен, выделенный из полипептида, выбранного из группы, состоящей из: FcRγ, FcRβ, CD3γ, CD3δ, CD3ε, CD3ζ, CD22, CD79a, CD79b и CD66d.
c. Рецепторы Daric
В частных вариантах сконструированные иммунные эффекторные клетки содержит один или более рецепторов Daric. Используемый в настоящей заявке термин «рецептор Daric» относится к многоцепочечному сконструированному антигенному рецептору. В одном варианте реализации Т-клетки сконструированы путем введения DSB в один или более генов PD-1 в присутствии донорной матрицы репарации, кодирующей один или более компонентов Daric. В частном варианте реализации Daric или один или более его компонентов встроен в DSB в одном гене PD-1.
В одном варианте реализации сконструированные Т-клетки содержат Daric, который не встроен в ген PD-1, и одно или более из следующего: демпфер иммуносупрессивного сигнала, флип-рецептор, альфа- и/или бета-цепь сконструированного Т-клеточного рецептора (TCR), химерный антигенный рецептор (CAR), или рецептор Daric или его компоненты встроены в DSB в одном или нескольких генах PD-1.
Иллюстративные примеры структур и компонентов Daric раскрыты в публикации РСТ №WO2015/017214 и публикации патента США №20150266973, каждая из которых включена в настоящую заявку посредством ссылки во всей своей полноте.
В одном варианте реализации донорная матрица репарации содержит следующие компоненты Daric: сигнальный полипептид, содержащий первый домен мультимеризации, первый трансмембранный домен и один или более внутриклеточных костимулирующих сигнальных доменов и/или первичных сигнальных доменов; и связывающий полипептид, содержащий связывающий домен, второй домен и мультимеризации, необязательно, второй трансмембранный домен. Функциональный Daric содержит мостиковый фактор, который способствует образованию рецепторного комплекса Daric на поверхности клетки, причем мостиковый фактор, ассоциирован с и расположен между доменами мультимеризации сигнального полипептида и связывающего полипептида.
В частных вариантах реализации первый и второй домены мультимеризации ассоциируются с мостиковым фактором, выбранным из группы, состоящей из: рапамицина или его рапалога, кумермицина или его производного, гиббереллина или его производного, абсцизовой кислоты (ABA) или ее производного, метотрексат или его производное, циклоспорина А или его производного, FKCsA или его производного, триметоприма (Tmp) - синтетического лиганда для FKBP (SLF) или его производного и любой их комбинации.
Иллюстративные примеры аналогов рапамицина (рапалогов) включают аналоги, раскрытые в патенте США №. 6649595, причем структуры этих рапалогов включены в настоящее описание в качестве ссылки во всей их полноте. В некоторых случаях мостиковым фактором является рапалог с существенно сниженным иммуносупрессивным эффектом по сравнению с рапамицином. «Существенно сниженный иммуносупрессивный эффект» относится к рапалогу, обладающему иммуносупрессивным эффектом по меньшей мере в 0,1-0,005 раза меньшим, чем иммуносупрессивный эффект, наблюдаемый или ожидаемый для эквимолярного количества рапамицина, измеряемого либо клинически, либо в соответствующем in vitro (например, ингибирование T-клеточной пролиферации) или in vivo суррогате иммунодепрессивной активности человека. В одном варианте реализации «существенно сниженный иммуносупрессивный эффект» относится к рапалогу, имеющему значение EC 50 в таком анализе in vitro, которое по меньшей мере в 10-250 раз превышает значение EC 50, наблюдаемое для рапамицина в том же анализе.
Другие иллюстративные примеры рапалогов включают, среди прочего, эверолимус, новолимус, пимекролимус, ридафоролимус, такролимус, темсиролимус, умиролимус и зотаролимус.
В некоторых случаях изобретения домены мультимеризации будут ассоциироваться с мостиковым фактором, являющимся рапамицином или его рапалогом. Например, первый и второй домены мультимеризации представляют собой пару, выбранную из FKBP и FRB. Домены FRB представляют собой полипептидные области (белковые «домены»), которые способны образовывать трехчастный комплекс с белком FKBP и рапамицином или его рапалогом. Домены FRB присутствуют во многих встречающихся в природе белках, включая белки mTOR (также упоминаемые в литературе как FRAP, RAPT1 или RAFT) человека и других видов; дрожжевые белки, включая Tor1 и Tor2; и гомолог Candida FRAP. Информация о нуклеотидных последовательностях, клонировании и других аспектах этих белков уже известна в данной области. Например, регистрационный номер последовательности белка для mTOR человека - номер доступа GenBank №L34075.1 (Brown et al., Nature 369:756, 1994).
Домены FRB, подходящие для применения в частных вариантах реализации, рассматриваемых в настоящей заявке, как правило содержат по меньшей мере от приблизительно 85 до приблизительно 100 аминокислотных остатков. В некоторых вариантах изобретения аминокислотная последовательность FRB для применения в слитых белках по настоящему изобретению будет содержать последовательность из 93 аминокислот от Ile-2021 до Lys-2113 и мутацию T2098L, основанную на аминокислотной последовательности GenBank, номер доступа L34075. 1. Домен FRB для использования в Daric, рассматриваемый в частных вариантах реализации, будет способен связываться с комплексом белка FKBP, связанного с рапамицином или его рапалогом. В некоторых случаях пептидная последовательность домена FRB содержит (а) встречающуюся в природе пептидную последовательность, охватывающую по меньшей мере указанную 93-аминокислотную область человеческого mTOR или соответствующие области гомологичных белков; (b) вариант встречающегося в природе FRB, в котором до приблизительно десяти аминокислот, или от приблизительно 1 до приблизительно 5 аминокислот, или от приблизительно 1 до приблизительно 3 аминокислот, или в некоторых вариантах реализации только одна аминокислота встречающегося в природе пептид была удалена, вставлена или замещена; или (c) пептид, кодируемый молекулой нуклеиновой кислоты, способной селективно гибридизоваться с молекулой ДНК, кодирующей встречающийся в природе домен FRB, или последовательностью ДНК, которая была бы способна, если бы не вырожденность генетического кода, избирательно гибридизоваться с молекулой ДНК, кодирующей встречающийся в природе домен FRB.
FKBP (FK506-связывающие белки) представляют собой цитозольные рецепторы макролидов, такие как FK506, FK520 и рапамицин, и являются высоко консервативными для всех биологических видов. FKBP представляют собой белки или белковые домены, которые способны связываться с рапамицином или его рапалогом и дополнительно образовывать трехчастный комплекс с FRB-содержащим белком или слитым белком. Домен FKBP может также упоминаться как «рапамицин-связывающий домен». Информация, касающаяся нуклеотидных последовательностей, клонирования и других аспектов различных видов FKBP, известна в данной области (см., например, Staendart et al., Nature 346: 671, 1990 (FKBP12 человека); Kay, Biochem. J. 314: 361, 1996). Гомологичные белки FKBP у других видов млекопитающих, у дрожжей и у других организмов также известны в данной области и могут быть использованы в слитых белках, раскрытых в настоящей заявке. Домен FKBP, рассматриваемый в частных вариантах реализации, будет способен связываться с рапамицином или его рапалогом и участвовать в трехчастном комплексе с FRB-содержащим белком (что может быть определено любыми способами, прямыми или косвенными, для обнаружения такого связывания).
Иллюстративные примеры доменов FKBP, подходящих для использования в Daric, рассматриваемых в частных вариантах реализации включают, среди прочего: встречающуюся в природе пептидную последовательность FKBP, предпочтительно выделенную из человеческого белка FKBP12 (номер в GenBank AAA58476.1) или пептидную последовательность, выделенную из него, из FKBP другого человека, из FKBP мыши или другого млекопитающего или из FKBP какого-либо другого животного, дрожжевого или грибкового происхождения; вариант встречающейся в природе последовательности FKBP, в которой до приблизительно десяти аминокислот, или от приблизительно 1 до приблизительно 5 аминокислот, или от приблизительно 1 до приблизительно 3 аминокислот, или в некоторых вариантах реализации только одна аминокислота встречающегося в природе пептида была удалена, вставлена или заменена; или пептидную последовательность, кодируемую молекулой нуклеиновой кислоты, способной селективно гибридизоваться с молекулой ДНК, кодирующей встречающийся в природе FKBP, или последовательностью ДНК, которая была бы способна, если бы не вырожденность генетического кода, избирательно гибридизоваться с молекулой ДНК, кодирующей встречающийся в природе домен FKBP.
Другие иллюстративные примеры пар доменов мультимеризации, подходящих для использования в Daric, рассматриваемых в частных вариантах реализации включают, среди прочего, домены из FKBP и FRB, FKBP и кальцинейрина, FKBP и циклофилина, FKBP и бактериального DHFR, кальцинейрина и циклофилина, PYL1 и ABI1 или GIB1 и GAI, или их варианты.
В других вариантах реализации анти-мостиковый фактор блокирует ассоциацию сигнального полипептида и связывающего полипептида с мостиковым фактором. Например, циклоспорин или FK506 могут быть использованы в качестве анти-мостиковых факторов для титрования рапамицина и, следовательно, для остановки передачи сигналов, поскольку связан только один мультимеризационный домен. В некоторых случаях анти-мостиковым фактором (например, циклоспорин, FK506) является иммуносупрессивный агент.Например, иммуносупрессивный анти-мостиковый фактор может использоваться для блокирования или минимизации функции компонентов Daric, рассматриваемых в частных вариантах реализации и в то же время ингибировать или блокировать нежелательный или патологический воспалительный ответ в клинических условиях.
В одном варианте реализации первый домен мультимеризации содержит FRB T2098L, второй домен мультимеризации содержит FKBP12, а мостиковым фактором является рапалог AP21967.
В другом варианте реализации первый домен мультимеризации содержит FRB, второй домен мультимеризации содержит FKBP12, а фактор связывания представляет собой рапамицин, темсиролимус или эверолимус.
В частных вариантах реализации сигнальный полипептид содержит первый трансмембранный домен и связывающий полипептид содержит второй трансмембранный домен или GPI-якорь. Иллюстративные примеры первого и второго трансмембранных доменов выделены из полипептида, независимо выбранного из группы, состоящей из: CD3δ, CD3ε, CD3γ, CD3ζ, CD4, CD5, CD8α, CD9, CD 16, CD22, CD27, CD28, CD33, CD37, CD45, CD64, CD80, CD86, CD 134, CD137, CD152, CD154, AMN и PD-1.
В одном варианте реализации сигнальный полипептид содержит один или более внутриклеточных костимулирующих сигнальных доменов и/или первичных сигнальных доменов.
Иллюстративные примеры первичных сигнальных доменов, подходящих для использования в компонентах передачи сигналов Daric, рассматриваемых в частных вариантах реализации включают в себя домены, которые получены из FcRγ, FcRβ, CD3γ, CD3δ, CD3ε, CD3ζ, CD22, CD79a, CD79b и CD66d. В частных предпочтительных вариантах реализации изобретения сигнальный компонент Daric содержит первичный сигнальный домен CD3 и один или более костимуляторных сигнальных доменов. Внутриклеточные первичные сигнальные и костимулирующие сигнальные домены могут быть связаны в любом порядке в тандеме с карбоксильным концом трансмембранного домена.
Иллюстративные примеры таких костимулирующих молекул, подходящих для использования в сигнальных компонентах Daric, рассматриваемых в частных вариантах реализации включают TLR1, TLR2, TLR3, TLR4, TLR5, TLR6, TLR7, TLR8, TLR9, TLR10, CARD11, CD2, CD7, CD27, CD28, CD30, CD40, CD54 (ICAM), CD83, CD134 (OX40), CD137 (4-1BB), CD278 (ICOS), DAP10, LAT, NKD2C, SLP76, TRIM и ZAP70. В одном варианте реализации сигнальный компонент Daric содержит один или более костимулирующих сигнальных доменов, выбранных из группы, состоящей из CD28, CD137 и CD134, и первичного сигнального домена CD3ζ.
В частных вариантах реализации Daric-связывающий компонент содержит связывающий домен. В одном варианте реализации такой связывающий домен представляет собой антитело или его антигенсвязывающий фрагмент.
Антитело или его антигенсвязывающий фрагмент содержит, по меньшей мере вариабельную область легкой или тяжелой цепи иммуноглобулина, которая специфически распознает и связывает эпитоп антигена-мишени, такого как пептид, липид, полисахарид или нуклеиновая кислота, содержащая антигенный детерминант, например, распознаваемые иммунной клеткой. Антитела включают антигенсвязывающие фрагменты, например, Camel Ig (антитело верблюдовых или его фрагмент VHH), Ig NAR, фрагменты Fab, фрагменты Fab', фрагменты F(ab)' 2, фрагменты F(ab)'3, Fv, одноцепочечный вариабельный фрагмент антитела Fv («scFv»), бис-scFv, (scFv) 2, миниантитело, диатело, триатело, тетратело, стабилизированный дисульфидом белок Fv («dsFv») и однодоменное антитело (sdAb, Nanobody) или другие соответствующие фрагменты антител. Термин также включает сконструированные формы, такие как химерные антитела (например, гуманизированные мышиные антитела), гетероконъюгатные антитела (такие как биспецифичные антитела) и их антигенсвязывающие фрагменты. См. также, Pierce Catalog and Handbook, 1994-1995 (Pierce Chemical Co., Rockford, IL); Kuby, J., Immunology, 3rd Ed., W. H. Freeman & Co., New York, 1997.
В одном предпочтительном варианте реализации связывающий домен представляет собой scFv.
В другом предпочтительном варианте реализации связывающий домен представляет собой антитело верблюдовых.
В частных вариантах реализации связывающий компонент Daric содержит внеклеточный домен, который связывает антиген, выбранный из группы, состоящей из: альфа-фолатного рецептора, 5T4, интегрина αvβ6, BCMA, B7-H3, B7-H6, CAIX, CD16, CD19, CD20, CD22, CD30, CD33, CD44, CD44v6, CD44v7/8, CD70, CD79a, CD79b, CD123, CD138, CD171, CEA, CSPG4, EGFR, семейства EGFR включая ErbB2 (HER2), EGFRvIII, EGP2, EGP40, EPCAM, EphA2, EpCAM, FAP, фетального AchR, FRα, GD2, GD3, глипикана-3 (GPC3), HLA-A1+MAGE1, HLA-A2+MAGE1, HLA-A3+MAGE1, HLA-A1+NY-ESO-1, HLA-A2+NY-ESO-1, HLA-A3+NY-ESO-1, IL-11Rα, IL-13Rα2, Лямбда, Lewis-Y, Каппа, мезотелина, Muc1, Muc16, NCAM, лигандов NKG2D, NY-ESO-1, PRAME, PSCA, PSMA, ROR1, SSX, сурвивина, TAG72, TEMs, VEGFR2 и WT-1.
В одном варианте реализации связывающий компонент Daric содержит внеклеточный домен, например, антитело или его антигенсвязывающий фрагмент, который связывает комплекс MHC-пептид, такой как комплекс MHC класса I-пептид или комплекс MHC класса II-пептид.
В частных вариантах реализации компоненты Daric, рассматриваемые в настоящей заявке, содержат линкер или спейсер, который соединяет два белка, полипептида, пептида, домена, области или мотива. В некоторых вариантах реализации линкер содержит от приблизительно двух до приблизительно 35 аминокислот, или от приблизительно четырех до приблизительно 20 аминокислот, или от приблизительно восьми до приблизительно 15 аминокислот, или от приблизительно 15 до приблизительно 25 аминокислот. В других вариантах реализации спейсер может иметь конкретную структуру, такую как домен антитела CH2CH3, шарнирный домен или тому подобное. В одном варианте реализации спейсер содержит домены CH2 и CH3 белков IgG1, IgG4 или IgD.
В частных вариантах реализации компоненты Daric, рассматриваемые в настоящей заявке, содержат один или более «шарнирных доменов», которые играют свою роль в позиционировании доменов для обеспечения надлежащего межклеточного контакта, связывания антигена и активации. Daric может содержать один или более шарнирных доменов между связывающим доменом и доменом мультимеризации и/или трансмембранным доменом (TM) или между доменом мультимеризации и трансмембранным доменом. Шарнирный домен может быть получен из природного, синтетического, полусинтетического или рекомбинантного источника. Шарнирный домен может включать аминокислотную последовательность встречающейся в природе шарнирной области иммуноглобулина или измененную шарнирную область иммуноглобулина. В частном варианте реализации шарнир представляет собой шарнир CD8α или шарнир CD4.
В одном варианте реализации изобретения Daric содержит сигнальный полипептид, содержащий первый домен мультимеризации FRB T2098L, трансмембранный домен CD8, костимуляторный домен 4-1BB и первичный сигнальный домен CD3ζ; связывающий полипептид содержит scFv, который связывает CD19, второй мультимеризационный домен FKBP12 и трансмембранный домен CD4; а мостиковым фактором является рапалог AP21967.
В одном варианте реализации изобретения Daric содержит сигнальный полипептид, содержащий первый мультимеризационный домен FRB, трансмембранный домен CD8, костимуляторный домен 4-1BB и первичный сигнальный домен CD3ζ; связывающий полипептид содержит scFv, который связывает CD19, второй мультимеризационный домен FKBP12 и трансмембранный домен CD4; а мостиковым фактором является рапамицин, темсиролимус или эверолимус.
d. Дзетакины
В частных вариантах реализации сконструированные иммунные эффекторные клетки, рассматриваемые в настоящей заявке, содержат один или более химерных рецепторов цитокинов. В одном варианте реализации Т-клетки сконструированы путем введения DSB в один или более генов PD-1 в присутствии донорной матрицы репарации, кодирующей CAR. В частном варианте реализации химерный цитокиновый рецептор встроен в DSB в одном гене PD-1.
В одном варианте реализации сконструированные T- клетки, рассматриваемые в настоящей заявке, содержат химерный рецептор цитокинов, который не встроен в ген PD-1 и один или более из: демпфера иммуносупрессивного сигнала, флип-рецептора, альфа- и/или бета-цепи сконструированного Т-клеточного рецептора (TCR), химерного антигенного рецептора (CAR), рецептора Daric или его компонентов, или химерный цитокиновый рецептор встроен в DSB в одном или нескольких генах PD-1.
В различных вариантах реализации Т-клетки с отредактированным геномом экспрессируют химерный цитокиновый рецептор, который перенаправляет цитотоксичность на опухолевые клетки. Дзетакины представляют собой химерные трансмембранные иммунорецепторы, которые содержат внеклеточный домен, содержащий растворимый рецепторный лиганд, связанный с вспомогательной областью, способной связывать внеклеточный домен с клеточной поверхностью, трансмембранной областью и внутриклеточным сигнальным доменом. Дзетакины, когда они экспрессируются на поверхности Т-лимфоцитов, направляют активность Т-клеток на те клетки, которые экспрессируют рецептор, для которого специфичен растворимый рецепторный лиганд. Химерные иммунорецепторы дзетакинов перенаправляют антиген-специфичность Т-клеток с возможностью применения для лечения различных видов рака, в частности, через аутокринные/паракринные цитокиновые системы, используемые злокачественными опухолями человека.
В частных вариантах реализации химерный цитокиновый рецептор содержит иммуносупрессивный цитокин или его связывающий цитокиновый рецептор вариант, линкер, трансмембранный домен и внутриклеточный сигнальный домен.
В частных вариантах реализации цитокин или его связывающий цитокиновый рецептор вариант выбирают из группы, состоящей из: интерлейкина-4 (IL-4), интерлейкина-6 (IL-6), интерлейкина-8 (IL-8), интерлейкина-10 (IL-10) и интерлейкина-13 (IL-13).
В некоторых вариантах реализации линкер содержит домен CH2CH3, шарнирный домен или тому подобное. В одном варианте реализации линкер содержит домены CH2 и CH3 IgG1, IgG4 или IgD. В одном варианте реализации линкер содержит шарнирный домен CD8α или CD4.
В частных вариантах реализации трансмембранный домен выбран из группы, состоящей из: альфа- или бета-цепи рецептора Т-клеток, CD3δ, CD3ε, CD3γ, CD3ζ, CD4, CD5, CD8α, CD9, CD 16, CD22, CD27, CD28, CD33, CD37, CD45, CD64, CD80, CD86, CD 134, CD137, CD152, CD154, AMN θ PD-1.
ITAM, содержащего первичный сигнальный домен и/или костимуляторный домен.
В частных вариантах реализации внутриклеточный сигнальный домен выбран из группы, состоящей из: FcRγ, FcRβ, CD3γ, CD3δ, CD3ε, CD3ζ, CD22, CD79a, CD79b и CD66d.
В частных вариантах реализации внутриклеточный сигнальный домен выбран из группы, состоящей из: TLR1, TLR2, TLR3, TLR4, TLR5, TLR6, TLR7, TLR8, TLR9, TLR10, CARD11, CD2, CD7, CD27, CD28, CD30, CD40, CD54 (ICAM), CD83, CD134 (OX40), CD137 (4-1BB), CD278 (ICOS), DAP10, LAT, NKD2C, SLP76, TRIM и ZAP70.
В одном варианте реализации химерный цитокиновый рецептор содержит один или более костимуляторных сигнальных доменов, выбранных из группы, состоящей из CD28, CD137 и CD134, и первичного сигнального домена CD3ζ.
D. Полипептиды
В настоящей заявке рассматриваются различные полипептиды, включая, среди прочего, варианты хоминг-эндонуклеазы, megaTAL и слитые полипептиды. В предпочтительных вариантах реализации полипептид содержит аминокислотную последовательность, представленную в SEQ ID NO: 1-24 и 60-64. «Полипептид», «полипептидный фрагмент», «пептид» и «белок» используются взаимозаменяемо, если не указано иное, и в соответствии с общепринятым значением, то есть как последовательность аминокислот.В одном варианте реализации «полипептид» включает слитые полипептиды и другие варианты. Полипептиды могут быть получены с использованием любой из множества хорошо известных рекомбинантных и/или синтетических методик. Полипептиды не ограничены конкретной длиной, например, они могут содержать полноразмерную белковую последовательность, фрагмент полноразмерного белка или слитый белок и могут включать посттрансляционные модификации полипептида, например гликозилирование, ацетилирование, фосфорилирование и тому подобное, а также другие модификации, известные в данной области техники, как встречающиеся в природе, так и не встречающиеся в природе.
Понятия «выделенный белок», «выделенный пептид» или «выделенный полипептид» и тому подобное, используемые в настоящей заявке, относятся к синтезу, выделению и/или очистке молекулы пептида или полипептида in vitro из клеточной среды и из ассоциации с другими компонентами клетки, т.е. она существенно не ассоциирована с веществами in vivo.
Иллюстративные примеры полипептидов, рассматриваемых в частных вариантах реализации включают, среди прочего, варианты хоминг-эндонуклеазы, megaTAL, изменяющие структуру концов нуклеазы, слитые полипептиды и их варианты.
Полипептиды включают в себя «варианты полипептидов». Варианты полипептидов могут отличаться от встречающихся в природе полипептидов заменами, делециями, добавлениями и/или инсерциями одной или нескольких аминокислот.Такие варианты могут встречаться в природе или могут быть получены синтетическим путем, например, путем модификации одной или нескольких аминокислот из вышеуказанных полипептидных последовательностей. Например, в частных вариантах реализации может быть желательно улучшить биологические свойства хоминг-эндонуклеазы, megaTAL или тому подобного, связывающих и расщепляющих сайт-мишень в гене PD-1 человека, путем введения одной или нескольких замен, делеции, добавления и/или инсерции в полипептид. В частных вариантах реализации полипептиды включают полипептиды, имеющие идентичность аминокислот по меньшей мере на приблизительно 65%, 70%, 71%, 72%, 73%, 74%, 75%, 76%, 77%, 78%, 79%, 80%, 81%, 82%, 83%, 84%,85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% с любой из эталонных последовательностей, рассматриваемых в настоящей заявке, обычно, когда указанный вариант сохраняет по меньшей мере один тип биологической активности эталонной последовательности.
Варианты полипептидов включают биологически активные «полипептидные фрагменты». Иллюстративные примеры биологически активных полипептидных фрагментов включают ДНК-связывающие домены, нуклеазные домены и тому подобное. Используемый в настоящей заявке термин «биологически активный фрагмент» или «минимально биологически активный фрагмент» относится к полипептидному фрагменту, который сохраняет по меньшей мере 100%, по меньшей мере 90%, по меньшей мере 80%, по меньшей мере 70%, по меньшей мере 60%, по меньшей мере 50%, по меньшей мере 40%, по меньшей мере 30%, по меньшей мере 20%, по меньшей мере 10% или, по меньшей мере 5% от встречающейся в природе полипептидной активности. В предпочтительных вариантах реализации биологическая активность представляет собой аффинность связывания и/или активность расщепления для последовательности-мишени. В некоторых вариантах реализации полипептидный фрагмент может содержать аминокислотную цепь длиной по меньшей мере от 5 до 1700 аминокислот.Понятно, что в некоторых вариантах реализации длина фрагментов не менее 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 55, 60, 65, 70, 75, 80, 85, 90, 95, 100, 110, 150, 200, 250, 300, 350, 400, 450, 500, 550, 600, 650, 700, 750, 800, 850, 900, 950, 1000, 1100, 1200, 1300, 1400, 1500, 1600, 1700 или более аминокислот.В частных вариантах реализации полипептид содержит биологически активный фрагмент варианта хоминг-эндонуклеазы. В частных вариантах реализации полипептиды, описанные в настоящей заявке, могут содержать одну или более аминокислот, обозначенных как «X». «X», если присутствует в аминокислоте SEQ ID NO, относится к любой аминокислоте. Один или более остатков «X» могут присутствовать на N- и C-концах аминокислотной последовательности, представленной в конкретных SEQ ID NO, рассматриваемых в настоящей заявке. Если аминокислоты «Х» отсутствуют, оставшаяся аминокислотная последовательность, представленная в SEQ ID NO, может считаться биологически активным фрагментом.
В частных вариантах реализации полипептид содержит биологически активный фрагмент варианта хоминг-эндонуклеазы, например, SEQ ID NO: 3-14 и 60-63, или megaTAL (SEQ ID NO: 15-23 и 64). Биологически активный фрагмент может содержать усечение N-конца и/или усечение C-конца. В частном варианте реализации биологически активный фрагмент не содержит или содержит делецию 1, 2, 3, 4, 5, 6, 7 или 8 N-концевых аминокислот варианта хоминг-эндонуклеазы по сравнению с последовательностью соответствующей хоминг-эндонуклеазы дикого типа, более предпочтительно - делецию 4 N-концевых аминокислот варианта хоминг-эндонуклеазы по сравнению с соответствующей последовательностью хоминг-эндонуклеазы дикого типа. В частном варианте реализации биологически активный фрагмент не содержит или содержит делецию 1, 2, 3, 4 или 5 С-концевых аминокислот варианта хоминг-эндонуклеазы по сравнению с соответствующей последовательностью хоминг-эндонуклеазы дикого типа, более предпочтительно делецию 2 C-концевых аминокислот варианта хоминг-эндонуклеазы по сравнению с соответствующей последовательностью хоминг-эндонуклеазы дикого типа. В частном предпочтительном варианте реализации биологически активный фрагмент не содержит или содержит делецию 4 N-концевых аминокислот и 2 C-концевых аминокислот варианта хоминг-эндонуклеазы по сравнению с соответствующей последовательностью хоминг-эндонуклеазы дикого типа.
В частном варианте реализации вариант I-OnuI содержит делецию 1, 2, 3, 4, 5, 6, 7 или 8 следующих N-концевых аминокислот: М, А, Y, М, S, R, R, E; и/или делецию следующих 1, 2, 3, 4 или 5 С-концевых аминокислот: R, G, S, F, V.
В частном варианте реализации вариант I-OnuI содержит делецию или замену 1, 2, 3, 4, 5, 6, 7 или 8 следующих N-концевых аминокислот: M, A, Y, M, S, R, R, E; и/или делецию или замену следующих 1, 2, 3, 4, или 5 C-концевых аминокислот: R, G, S, F, V.
В частном варианте реализации вариант I-OnuI содержит делецию 1, 2, 3, 4, 5, 6, 7 или 8 следующих N-концевых аминокислот: М, А, Y, М, S, R, R, E; и/или делецию следующих 1 или 2 С-концевых аминокислот: F, V.
В частном варианте реализации вариант I-OnuI содержит делецию или замену 1, 2, 3, 4, 5, 6, 7, или 8 следующих N-концевых аминокислот: M, A, Y, M, S, R, R, E; и/или делецию или замену следующих 1 или 2 С-концевых аминокислот: F, V.
Как отмечено выше, полипептиды могут быть изменены различными способами, включая замены, делеции, усечения и инсерции аминокислот.Способы таких манипуляций как правило известны в данной области. Например, варианты аминокислотной последовательности эталонного полипептида могут быть получены мутациями в ДНК. Методы мутагенеза и изменения нуклеотидных последовательностей хорошо известны в данной области. См. например, Kunkel (1985, Proc. Natl. Acad. Sci. USA. 82: 488-492), Kunkel et al., (1987, Methods in Enzymol, 154: 367-382), Патент США №4873192, Watson, J. D. et al., (Molecular Biology of the Gene, Fourth Edition, Benjamin/Cummings, Menlo Park, Calif., 1987) и ссылках, приведенных там. Руководство по подходящим аминокислотным заменам, которые не влияют на биологическую активность интересующего белка, можно найти в модели Dayhoff et al., (1978) Atlas of Protein Sequence and Structure (Natl. Biomed. Res. Found., Washington, D.C.).
В некоторых вариантах реализации вариант будет содержать одну или более консервативных замен. «Консервативная замена» представляет собой замену аминокислоты другой аминокислотой, которая обладает сходными свойствами, так что специалист в области химии пептидов может ожидать, что вторичная структура и гидропатическая природа полипептида практически не изменятся. Модификации могут быть внесены в структуру полинуклеотидов и полипептидов, рассматриваемых в частных вариантах, причем полипептиды включают полипептиды, имеющие, по меньшей мере приблизительно и при этом получать функциональные молекулы, которые кодируют вариант или производное полипептида с желаемыми характеристиками. Когда желательно изменить аминокислотную последовательность полипептида для создания эквивалентного или даже улучшенного варианта полипептида, специалист в данной области может, например, изменить один или более кодонов кодирующей последовательности ДНК, например в соответствии с таблицей 1.
Таблица 1 - Аминокислотные кодоны
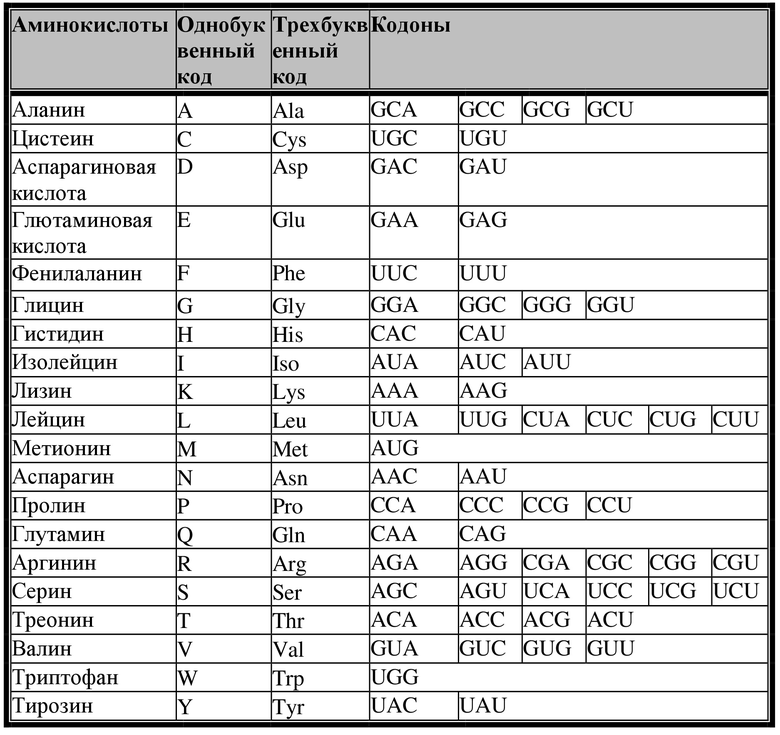 Руководство по определению того, какие именно аминокислотные остатки могут быть замещены, вставлены или удалены без потери биологической активности, можно найти с помощью компьютерных программ, хорошо известных в данной области, таких как программное обеспечение DNASTAR, DNA Strider, Geneious, Mac Vector или Vector NTI. Предпочтительно аминокислотные замены в вариантах белка, раскрытых в данном описании, представляют собой консервативные аминокислотные замены, то есть замены одинаково заряженных или незаряженных аминокислот.Консервативная замена аминокислот включает замену одной из семейства аминокислот, которые родственны в их боковых цепях. Встречающиеся в природе аминокислоты, как правило, делятся на четыре семейства: кислые (аспартат, глутамат), основные (лизин, аргинин, гистидин), неполярные (аланин, валин, лейцин, изолейцин, пролин, фенилаланин, метионин, триптофан) и незаряженные полярные (глицин, аспарагин, глутамин, цистеин, серин, треонин, тирозин) аминокислоты. Фенилаланин, триптофан и тирозин иногда классифицируют как ароматические аминокислоты. В пептиде или белке подходящие консервативные замены аминокислот известны специалистам в данной области и обычно могут быть сделаны без изменения биологической активности получающейся в результате молекулы. Специалисты в данной области признают, что, как правило, единичные аминокислотные замены в несущественных областях полипептида существенно не изменяют биологическую активность (см., например, Watson et al. Molecular Biology of the Gene, 4th Edition, 1987, The Benjamin/Cummings Pub. Co., p.224).
Руководство по определению того, какие именно аминокислотные остатки могут быть замещены, вставлены или удалены без потери биологической активности, можно найти с помощью компьютерных программ, хорошо известных в данной области, таких как программное обеспечение DNASTAR, DNA Strider, Geneious, Mac Vector или Vector NTI. Предпочтительно аминокислотные замены в вариантах белка, раскрытых в данном описании, представляют собой консервативные аминокислотные замены, то есть замены одинаково заряженных или незаряженных аминокислот.Консервативная замена аминокислот включает замену одной из семейства аминокислот, которые родственны в их боковых цепях. Встречающиеся в природе аминокислоты, как правило, делятся на четыре семейства: кислые (аспартат, глутамат), основные (лизин, аргинин, гистидин), неполярные (аланин, валин, лейцин, изолейцин, пролин, фенилаланин, метионин, триптофан) и незаряженные полярные (глицин, аспарагин, глутамин, цистеин, серин, треонин, тирозин) аминокислоты. Фенилаланин, триптофан и тирозин иногда классифицируют как ароматические аминокислоты. В пептиде или белке подходящие консервативные замены аминокислот известны специалистам в данной области и обычно могут быть сделаны без изменения биологической активности получающейся в результате молекулы. Специалисты в данной области признают, что, как правило, единичные аминокислотные замены в несущественных областях полипептида существенно не изменяют биологическую активность (см., например, Watson et al. Molecular Biology of the Gene, 4th Edition, 1987, The Benjamin/Cummings Pub. Co., p.224).
В одном варианте реализации, где желательна экспрессия двух или более полипептидов, полинуклеотидные последовательности, кодирующие их, могут быть разделены последовательностью IRES, как описано в другом месте в настоящей заявке.
Полипептиды, рассматриваемые в частных вариантах реализации включают слитые полипептиды. В частных вариантах реализации предлагаются слитые полипептиды и полинуклеотиды, кодирующие слитые полипептиды. Слитые полипептиды и слитые белки обозначают полипептид, имеющий по меньшей мере два, три, четыре, пять, шесть, семь, восемь, девять или десять полипептидных сегментов.
В другом варианте реализации два или более полипептида могут быть экспрессированы в виде слитого белка, который содержит одну или более последовательностей саморасщепляемых полипептидов, как описано в другом месте в настоящей заявке.
В одном варианте реализации рассматриваемый в настоящей заявке слитый белок содержит один или более ДНК-связывающих доменов и одну или более нуклеаз, а также один или более линкеров и/или саморасщепляемых полипептидов.
В одном варианте реализации рассматриваемый в настоящей заявке слитый белок содержит вариант нуклеазы; линкер или саморасщепляемый пептид; и фермент, изменяющий участки концов, включая, среди прочего, 5'-3' экзонуклеазу, 5'-3' щелочную экзонуклеазу и 3'-5' экзонуклеазу (например, Trex2).
Слитые полипептиды могут содержать один или более полипептидных доменов или сегментов, включая, среди прочего, сигнальные пептиды, проникающие в клетку пептидные домены (CPP), ДНК-связывающие домены, нуклеазные домены и т.д., метки эпитопа (например, мальтозосвязывающий белок («MBP»), глутатион-S-трансферазу (GST), HIS6, MYC, FLAG, V5, VSV-G и HA), полипептидные линкеры и сигналы расщепления полипептидов. Слитые полипептиды обычно связаны С-концом с N-концом, хотя они также могут быть связаны С-концом с С-концом, N-концом с N-концом или N-концом с С-концом. В частных вариантах реализации полипептиды слитого белка могут быть в любом порядке. Слитые полипептиды или слитые белки могут также включать консервативно модифицированные варианты, полиморфные варианты, аллели, мутанты, подпоследовательности и межвидовые гомологи, при условии, что желаемая активность слитого полипептида сохраняется. Слитые полипептиды могут быть получены методами химического синтеза или химической связью между двумя фрагментами или, как правило, могут быть получены с использованием других стандартных методик. Лигированные последовательности ДНК, содержащие слитый полипептид, функционально связаны с подходящими элементами контроля транскрипции или трансляции, как описано в другом месте в настоящей заявке.
Слитые полипептиды могут необязательно содержать линкер, который можно использовать для связывания одного или нескольких полипептидов или доменов внутри полипептида. Последовательность пептидного линкера может быть использована для разделения любых двух или более полипептидных компонентов на расстояние, достаточное для того, чтобы каждый полипептид складывался в свои соответствующие вторичные и третичные структуры так, чтобы позволить полипептидным доменам выполнять свои желаемые функции. Такая пептидная линкерная последовательность включается в слитый полипептид с использованием стандартных методик в данной области. Подходящие пептидные линкерные последовательности могут быть выбраны на основании следующих факторов: (1) их способности принимать гибкую расширенную конформацию; (2) их неспособности принимать вторичную структуру, которая могла бы взаимодействовать с функциональными эпитопами на первом и втором полипептидах; и (3) отсутствия гидрофобных или заряженных остатков, которые могли бы реагировать с полипептидными функциональными эпитопами. Предпочтительные пептидные линкерные последовательности содержат остатки Gly, Asn и Ser. Другие почти нейтральные аминокислоты, такие как Thr и Ala, также могут быть использованы в линкерной последовательности. Аминокислотные последовательности, которые можно эффективно использовать в качестве линкеров, включают последовательности, раскрытые в Maratea et al., Gene 40:39-46, 1985; Murphy et al., Proc. Natl. Acad. Sci. USA 83:8258-8262, 1986; Патент США №4,935,233 и Патент США №4,751,180. Последовательности линкеров не требуются, когда частный сегмент полипептида слияния содержит несущественные N-концевые аминокислотные области, которые можно использовать для разделения функциональных доменов и предотвращения стерического влияния. Предпочтительными линкерами обычно являются гибкие аминокислотные подпоследовательности, которые синтезируются как часть рекомбинантного слитого белка. Линкерные полипептиды могут иметь длину от 1 до 200 аминокислот, длину от 1 до 100 аминокислот или длину от 1 до 50 аминокислот, включая все целые значения между ними.
Типичные линкеры включают, среди прочего, следующие аминокислотные последовательности: глициновые полимеры (G)n; глицин-сериновые полимеры (G1-5S1-5)n, где n представляет собой целое число, величиной по меньшей мере один, два, три, четыре, или пять; глицин-аланиновые полимеры; аланин-сериновые полимеры; GGG (SEQ ID NO: 69); DGGGS (SEQ ID NO: 70); TGEKP (SEQ ID NO: 71) (см. например, Liu et al., PNAS 5525-5530 (1997)); GGRR (SEQ ID NO: 72) (Pomerantz et al. 1995, supra); (GGGGS)n где n=1, 2, 3, 4 или 5 (SEQ ID NO: 73) (Kim et al., PNAS 93, 1156-1160 (1996.); EGKSSGSGSESKVD (SEQ ID NO: 74) (Chaudhary et al., 1990, Proc. Natl. Acad. Sci. U.S.A. 87:1066-1070); KESGSVSSEQLAQFRSLD (SEQ ID NO: 75) (Bird et al., 1988, Science 242:423-426), GGRRGGGS (SEQ ID NO: 76); LRQRDGERP (SEQ ID NO: 77); LRQKDGGGSERP (SEQ ID NO: 78); LRQKD(GGGS)2ERP (SEQ ID NO: 79). В альтернативном варианте гибкие линкеры могут быть рационально разработаны с использованием компьютерной программы, способной моделировать как сайты связывания ДНК, так и сами пептиды (Desjarlais & Berg, PNAS 90:2256-2260 (1993), PNAS 91:11099-11103 (1994) или методами фагового дисплея.
Слитые полипептиды могут дополнительно содержать сигнал расщепления полипептида между каждым из полипептидных доменов, описанных в настоящей заявке, или между эндогенной открытой рамкой считывания и полипептидом, кодируемым донорной матрицей репарации. Кроме того, сайт расщепления полипептида может быть введен в любую линкерную пептидную последовательность. Типичные сигналы расщепления полипептидов включают сайты распознавания расщепления полипептидов, такие как сайты расщепления протеазами, сайты расщепления нуклеазами (например, сайты распознавания редких ферментов рестрикции, сайты распознавания саморасщепляемого рибозима) и саморасщепляемые вирусные олигопептиды (см. deFelipe and Ryan, 2004. Traffic, 5(8); 616-26).
Подходящие сайты расщепления протеазами и саморасщепляемый пептид известны специалистам в данной области (см., Например, в Ryan et al., 1997. J. Gener. Virol. 78, 699-722; Scymczak et al. (2004) Nature Biotech. 5, 589-594). Типичные сайты расщепления протеазами включают, среди прочего, сайты расщепления протеазами NIa потивируса (например, протеазы вируса гравировки табака), протеазами потивируса HC, протеазами потивируса P1 (P35), протеазами биовируса NIa, протеазами, кодируемыми биовирусом RNA-2, протеазами афтовируса L, протеазами энтеровируса 2A, протеазами риновируса 2A, протеазами пикорнавируса 3C, протеазами комовируса 24K, протеазами неповируса 24K, 3C-подобной протеазой RTSV (сферический вирус риса тунгро), 3C-подобной протеазой PYVF (вирус желтой пятнистости пастернака), гепарином, тромбином, фактором Ха и энтерокиназой. Из-за высокой строгости расщепления сайты расщепления протеазой TEV (вирус гравировки табака) являются предпочтительными в одном варианте реализации, например, EXXYXQ (G/S) (SEQ ID NO: 80), например, ENLYFQG (SEQ ID NO: 81) и ENLYFQS (SEQ ID NO: 82), где X представляет собой любую аминокислоту (расщепление с помощью TEV происходит между Q и G или Q и S).
В некоторых вариантах реализации сайт саморасщепляемого полипептида содержит 2A или 2A-подобный сайт, последовательность или домен (Donnelly et al., 2001. J. Gen. Virol. 82:1027-1041). В частном варианте реализации вирусный пептид 2А представляет собой пептид афтовируса 2А, пептид потивируса 2А или пептид кардиовируса 2А.
В одном варианте реализации вирусный 2A-пептид выбран из группы, состоящей из: 2A-пептида вируса ящура (FMDV), 2A-пептида вируса ринита A (ERAV), 2A-пептида вируса Thosea asigna (TaV), 2A-пептида свиного тешовируса-1 (PTV-1), 2A-пептида тейловируса и 2A-пептида вируса энцефаломиокардита.
Иллюстративные примеры сайтов 2А представлены в таблице 2.
ТАБЛИЦА 2: Типичные сайты 2А включают следующие последовательности:

E. Полинуклеотиды
В частных вариантах реализации предусмотрены полинуклеотиды, кодирующие один или более вариантов хоминг-эндонуклеазы, megaTAL, ферменты, изменяющие структуру концов и слитые полипептиды, рассматриваемые в настоящей заявке. Используемые в настоящей заявке термины «полинуклеотид» или «нуклеиновая кислота» относятся к дезоксирибонуклеиновой кислоте (ДНК), рибонуклеиновой кислоте (РНК) и гибридам ДНК/РНК. Полинуклеотиды могут быть одноцепочечными или двухцепочечными, а также рекомбинантными, синтетическими или выделенными. Полинуклеотиды включают, среди прочего: предшественник мРНК (пре-мРНК), матричную РНК (мРНК), РНК, короткую интерферирующую РНК (киРНК), короткую шпилечную РНК (кшРНК), микроРНК (миРНК), рибозимы, геномную РНК (гРНК), РНК «плюс-цепь» (РНК (+)), РНК «минус-цепь» (РНК (-)), tracrРНК, crРНК, одиночную направляющую РНК (sgРНК), синтетическую РНК, синтетическую мРНК, геномную ДНК (гДНК), амплифицированную ПЦР ДНК, комплементарную ДНК (кДНК), синтетическую ДНК или рекомбинантную ДНК. Термин «полинуклеотиды» относитс к полимерной форме нуклеотидов длиной по меньшей мере 5, по меньшей мере 10, по меньшей мере 15, по меньшей мере 20, по меньшей мере 25, по меньшей мере 30, по меньшей мере 40, по меньшей мере 50, по меньшей мере 100, по меньшей мере 200, по меньшей мере 300, по меньшей мере 400, по меньшей мере 500, по меньшей мере 1000, по меньшей мере 5000, по меньшей мере 10000 или по меньшей мере 15000 или более нуклеотидов, либо рибонуклеотидов или дезоксирибонуклеотидов, либо модифицированных форм нуклеотидов любого типа, а также все промежуточные длины. Понятно, что «промежуточные длины» в данном контексте означают любую длину между указанными значениями, такую как 6, 7, 8, 9, и т.д.., 101, 102, 103, и т.д.; 151, 152, 153, и т.д.; 201, 202, 203, и т.д. В частных вариантах реализации полинуклеотиды или варианты имеют последовательность, идентичную на по меньшей мере или приблизительно 50%, 55%, 60%, 65%, 70%, 71%, 72%, 73%, 74%, 75%, 76%, 77%, 78%, 79%, 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% с эталонной последовательностью.
В частных вариантах реализации полинуклеотиды могут быть кодон-оптимизированными. Используемый в настоящей заявке термин «кодон-оптимизированный» относится к замене кодонов в полинуклеотиде, кодирующем полипептид, для увеличения экспрессии, стабильности и/или активности полипептида. Факторы, которые влияют на оптимизацию кодонов, включают, среди прочего, один или более из следующих: (i) вариация смещения кодонов между двумя или более организмами или генами или синтетически сконструированными таблицами смещения, (ii) вариация степени смещения кодонов в пределах организма, гена или набора генов, (iii) систематическая вариация кодонов, включая контекст, (iv) вариация кодонов в соответствии с их декодирующими тРНК, (v) вариация кодонов в соответствии с GC%, либо в целом, либо в одной позиции триплета, (vi) изменение степени сходства с эталонной последовательностью, например, встречающейся в природе последовательностью, (vii) вариация порогового значения частоты кодона, (viii) структурные свойства мРНК, транскрибируемых с последовательности ДНК, (ix) предшествующее знание о функции последовательностей ДНК, на котором должен строиться дизайн набора замещения кодонов, (x) систематическая вариация наборов кодонов для каждой аминокислоты и/или (xi) изолированное удаление ложных сайтов инициации трансляции.
Используемый в настоящей заявке термин «нуклеотид» относится к гетероциклическому азотистому основанию в N-гликозидной связи с фосфорилированным сахаром. Понятно, что нуклеотиды включают в себя природные основания и широкий спектр принятых в данной области модифицированных оснований. Такие основания обычно расположены в положении 1' нуклеотидной сахарной группы. Нуклеотиды как правило содержат основание, сахар и фосфатную группу. В рибонуклеиновой кислоте (РНК) сахар представляет собой рибозу, а в дезоксирибонуклеиновой кислоте (ДНК) сахар представляет собой дезоксирибозу, то есть сахар, в котором отсутствует гидроксильная группа, присутствующая в рибозе. Типичные природные азотистые основания включают пурины, аденозин (A) и гуанидин (G), а также пиримидины, цитидин (C) и тимидин (T) (или в контексте РНК, урацил (U)). Атом С-1 дезоксирибозы связан с N-1 пиримидина или N-9 пурина. Нуклеотиды обычно являются моно-, ди- или трифосфатми. Нуклеотиды могут быть немодифицированными или модифицированными по сахарной, фосфатной и/или основной группе (также взаимозаменяемо именуемые «нуклеотидные аналоги», «нуклеотидные производные», «модифицированные нуклеотиды», «неприродные нуклеотиды» и «нестандартные нуклеотиды»; см., например, WO 92)./07065 и WO 93/15187). Примеры модифицированных оснований нуклеиновых кислот обобщены Limbach et al., (1994, Nucleic Acids Res. 22, 2183-2196).
Нуклеотид также может рассматриваться как фосфатный эфир нуклеозида, причем этерификация происходит на гидроксильной группе, присоединенной к С-5 сахара. Используемый в настоящей заявке термин «нуклеозид» относится к гетероциклическому азотистому основанию в N-гликозидной связи с сахаром. В данной области техники известно, что нуклеозиды включают природные основания, а также включают хорошо известные модифицированные основания. Такие основания обычно располагаются в положении 1' нуклеозидного сахарного фрагмента. Нуклеозиды обычно содержат основание и сахарную группу. Нуклеозиды могут быть немодифицированными или модифицированными по сахарной и/или основной группе (взаимозаменяемо именуемые «нуклеозидные аналоги», «производные нуклеозидов», «модифицированные нуклеозиды», «неприродные нуклеозиды» или «нестандартные нуклеозиды»). Как также отмечено выше, примеры модифицированных оснований нуклеиновых кислот обобщены Limbach et al., (1994, Nucleic Acids Res. 22, 2183-2196).
Иллюстративные примеры полинуклеотидов включают, среди прочего, полинуклеотиды, кодирующие SEQ ID NO: 1-24 и 60-64, и полинуклеотидные последовательности, представленные в SEQ ID NO: 25-59 и 65-68.
В различных иллюстративных вариантах реализации, полинуклеотиды, рассматриваемые в настоящей заявке, включают, среди прочего, полинуклеотиды, кодирующие варианты хоминг-эндонуклеазы, megaTAL, ферменты, изменяющие структуру концов, слитые полипептиды, а также векторы экспрессии, вирусные векторы и транспортные плазмиды, содержащие полинуклеотиды, рассматриваемые в настоящей заявке.
Используемые в настоящей заявке термины «вариант полинуклеотида» и «вариант» и тому подобное относятся к полинуклеотидам, демонстрирующим существенную идентичность последовательности с эталонной полинуклеотидной последовательностью, или полинуклеотидам, которые гибридизуются с эталонной последовательностью в строгих условиях, которые определены ниже. Эти термины также охватывают полинуклеотиды, которые отличаются от эталонного полинуклеотида добавлением, делецией, заменой или модификацией по меньшей мере одного нуклеотида. Соответственно, термины «вариант полинуклеотида» и «вариант» включают полинуклеотиды, в которых один или более нуклеотидов были добавлены или удалены, или модифицированы, или заменены другими нуклеотидами. В связи с этим в данной области техники хорошо известно, что в отношении эталонного полинуклеотида могут быть сделаны определенные изменения, включая мутации, добавления, делеции и замены, при которых измененный полинуклеотид сохраняет биологическую функцию или активность эталонного полинуклеотида.
В одном варианте реализации полинуклеотид содержит нуклеотидную последовательность, которая гибридизуется с нуклеотидной последовательностью-мишенью в жестких условиях. Гибридизация в «жестких условиях» описывает протоколы гибридизации, в которых нуклеотидные последовательности, по меньшей мере на 60% идентичные друг другу, остаются гибридизованными. Обычно жесткие условия выбираются так, чтобы они были приблизительно на 5°C ниже, чем температура плавления (Tm) для конкретной последовательности при определенной ионной силе и pH. Tm - это температура (при определенной ионной силе, pH и концентрации нуклеиновой кислоты), при которой 50% зондов, комплементарных последовательности-мишени, гибридизуются с последовательностью-мишенью в равновесном состоянии. Поскольку последовательности-мишени обычно присутствуют в избытке, при Tm 50% зондов занято в равновесном состоянии.
Используемые в настоящей заявке выражения «идентичность последовательности» или, например, «содержать последовательность, идентичную на 50%», относятся к той степени, в которой последовательности идентичны на основании сравнения каждого нуклеотида или на основании сравнения каждой аминокислоты в окне сравнения. Таким образом, «процент идентичности последовательности» может быть рассчитан путем сравнения двух оптимально выровненных последовательностей в окне сравнения, определения количества положений, в которых идентичное основание нуклеиновой кислоты (например, A, T, C, G, I) или идентичный аминокислотный остаток (например, Ala, Pro, Ser, Thr, Gly, Val, Leu, Ile, Phe, Tyr, Trp, Lys, Arg, His, Asp, Glu, Asn, Gln, Cys и Met) встречаются в обеих последовательностях, для получения количества совпадающих позиций, с последующим делением количества совпадающих позиций на общее количество позиций в окне сравнения (т.е. размер окна) и умножением результата на 100, чтобы получить процент идентичности последовательности. Включены нуклеотиды и полипептиды, имеющие идентичность последовательности по меньшей мере приблизительно 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% или 100% с любой из эталонных последовательностей, описанных в настоящей заявке, при этом как правило, вариант полипептида сохраняет по меньшей мере одну биологическую активность эталонного полипептида.
Термины, используемые для описания родственности последовательностей между двумя или более полинуклеотидами или полипептидами, включают «эталонную последовательность», «окно сравнения», «идентичность последовательности», «процент идентичности последовательности» и «существенная идентичность». «Эталонная последовательность» имеет длину по меньшей мере 12, но часто от 15 до 18 и часто по меньшей мере 25 мономерных единиц, включая нуклеотиды и аминокислотные остатки. Поскольку каждый из двух полинуклеотидов может содержать (1) последовательность (т.е. только часть полной полинуклеотидной последовательности), которая является сходной между двумя полинуклеотидами, и (2) последовательность, которая расходится между двумя полинуклеотидами, сравнения последовательностей между двумя (или более) полинуклеотидами обычно выполняются путем сравнения последовательностей двух полинуклеотидов в «окне сравнения» для выявления и сравнения локальных областей сходства последовательностей. Термин «окно сравнения» относится к концептуальному сегменту из по меньшей мере 6 смежных позиций, обычно от приблизительно 50 до приблизительно 100, более обычно от приблизительно 100 до приблизительно 150, в котором последовательность сравнивается с эталонной последовательностью из того же числа смежных позиций после того, как эти две последовательности оптимально выровнены. Окно сравнения может содержать добавления или делеции (то есть, пропуски) величиной приблизительно 20% или меньше по сравнению с эталонной последовательностью (которая не содержит добавлений или делеций) для оптимального выравнивания двух последовательностей. Оптимальное выравнивание последовательностей для выравнивания окна сравнения может быть выполнено с помощью компьютеризированных реализаций алгоритмов (GAP, BESTFIT, FASTA и TFASTA в программе Wisconsin Genetics Software Package Release 7.0, Genetics Computer Group, 575 Science Drive Madison, Висконсин, США) или визуального сравнения и наилучшего выравнивания (т.е. приводящего к наибольшей процентной гомологии в окне сравнения), полученных любым из различных выбранных методов. Также можно сослаться на семейство программ BLAST, как, например, описано Altschul et al., 1997, Nucl. Acids Res. 25:3389. Подробное обсуждение анализа последовательностей можно найти в разделе 19.3 руководства Ausubel et al., Current Protocols in Molecular Biology, John Wiley & Sons Inc., 1994-1998, Chapter 15.
Термин «выделенный полинуклеотид», используемый в настоящей заявке, относится к полинуклеотиду, который был очищен от последовательностей, фланкирующих его в естественном состоянии, например фрагмент ДНК, который был отдален из последовательностей, которые обычно соседствуют с этим фрагментом. В частных вариантах реализации термин «выделенный полинуклеотид» относится к комплементарной ДНК (кДНК), рекомбинантному полинуклеотиду, синтетическому полинуклеотиду или другому полинуклеотиду, который не существует в природе и который был создан рукой человека.
В различных вариантах реализации полинуклеотид содержит мРНК, кодирующую полипептид, рассматриваемый в настоящей заявке, включая, среди прочего, вариант хоминг-эндонуклеазы, megaTAL и фермент, изменяющий структуру концов. В некоторых случаях мРНК содержит кэп, один или более нуклеотидов и поли(А) хвост.
Используемые в настоящей заявке термины «5' кэп» или «5' кэп-структура» или «5' кэп фрагмент» относятся к химической модификации, которая была встроена в 5'-конец мРНК. Кэп 5' участвует в ядерном экспорте, стабильности мРНК и трансляции.
В частных вариантах реализации рассматриваемая в настоящей заявке мРНК содержит 5'- кэп, содержащий 5'-ppp-5'-трифосфатную связь между концевым остатком гуанозинового кэпа и 5'-концевым транскрибированным смысловым нуклеотидом молекулы мРНК. Указанный 5'-гуанилатный кэп затем может быть метилирован с образованием остатка N7-метилгуанилата.
Иллюстративные примеры 5'- кэпа, подходящего для использования в частных вариантах реализации полинуклеотидов мРНК, рассматриваемых в настоящей заявке, включают, среди прочего: неметилированные аналоги 5'-кэпа, например, G(5')ppp(5')G, G(5')ppp(5')C, G(5')ppp(5')A; метилированные аналоги 5'-кэпа, например, m7G(5')ppp(5')G, m7G(5')ppp(5')C и m7G(5')ppp(5')A; диметилированные аналоги 5'-кэпа, например, m2,7G(5')ppp(5')G, m2,7G(5')ppp(5')C и m2,7G(5')ppp(5')A; триметилированные аналоги 5'-кэпа, например, m2,2,7G(5')ppp(5')G, m2,2,7G(5')ppp(5')C и m2,2,7G(5')ppp(5')A; диметилированные симметричные аналоги 5'-кэпа, например, m7G(5')pppm7(5')G, m7G(5')pppm7(5')C и m7G(5')pppm7(5')A; и анти-обратные аналоги 5'-кэпа, например, кэп Anti-Reverse Cap Analog (ARCA), специализированные 3'O-Me-m7G(5')ppp(5')G, 2'O-Me-m7G(5')ppp(5')G, 2'O-Me-m7G(5')ppp(5')C, 2'O-Me-m7G(5')ppp(5')A, m72'd(5')ppp(5')G, m72'd(5')ppp(5')C, m72'd(5')ppp(5')A, 3'O-Me-m7G(5')ppp(5')C, 3'O-Me-m7G(5')ppp(5')A, m73'd(5')ppp(5')G, m73'd(5')ppp(5')C, m73'd(5')ppp(5')A и их тетрафосфатные производные) (см., например, Jemielity et al., RNA, 9: 1108-1122 (2003)).
В частных вариантах реализации мРНК содержат 5'-кэп, который представляет собой 7-метилгуанилат («m7G») связаны через трифосфатный мостик с 5'-концом первого транскрибированного нуклеотида, что дает m7G(5')ppp(5')N, где N - любой нуклеозид.
В некоторых вариантах реализации мРНК содержат 5'- кэп, где кэп представляет собой структуру Cap0 (структуры Cap0 не имеют 2'-O-метильного остатка рибозы, присоединенной к основаниям 1 и 2), структуру Cap1 (структуры Cap1 имеют 2' -О-метильный остаток у основания 2) или структура Cap2 (структуры Cap2 имеют 2'-O-метильный остаток, присоединенный к обоим основаниям 2 и 3).
В одном варианте реализации мРНК содержит кэп m7G(5')ppp(5')G.
В одном варианте реализации мРНК содержит кэп ARCA.
В частных вариантах реализации рассматриваемая в настоящей заявке мРНК содержит один или более модифицированных нуклеозидов.
В одном варианте реализации мРНК содержит один или более модифицированных нуклеозидов, выбранных из группы, состоящей из следующих: псевдоуридин, пиридин-4-он рибонуклеозид, 5-азауридин, 2-тио-5-азауридин, 2-тиоуридин, 4-тио-псевдоуридин, 2-тио-псевдоуридин, 5-гидроксиуридин, 3-метилуридин, 5 -карбоксиметил-уридин, 1-карбоксиметил-псевдоуридин, 5-пропинил-уридин, 1-пропинил-псевдоуридин, 5-тауринометилуридин, 1-тауринометил-псевдоуридин, 5-тауринометил-2-тио-уридин, 1-таурино метил-4-тиоуридин, 5-метил-уридин, 1-метил-псевдоуридин, 4-тио-1-метил-псевдоуридин, 2-тио-1-метил-псевдоуридин, 1-метил-1-деаза-псевдоуридин, 2-тио-1-метил-1-деаза-псевдоуридин, дигидроуридин, дигидропсевдуридин, 2-тио-дигидроуридин, 2-тио-дигидропсевдуридин, 2-метоксиуридин, 2-метокси-4-тиоуридин, 4-метокси-псевдоуридин, 4-метокси-2-тио-псевдоуридин, 5-азацитидин, псевдоизоцитидин, 3-метилцитидин, N4-ацетилцитидин, 5-формилцитидин, N4-метилцитидин, 5-гидроксиметилцитидин, 1-метил-псевдоизоизитидин, пирроло-цитидин, пирроло-псевдоизоцитидин, 2-тиоцитидин, 2-тио-5-метилцитидин, 4-тио-псевдоизоцитидин, 4-тио-1-метил-псевдоизоцитидин, 4-тио-1-метил-1-деаза-псевдоизоцитидин, 1-метил-1-деаза-псевдоизоцитидин, зебуларин, 5-аза-зебуларин, 5-метилзебуларин, 5-аза-2-тио-зебуларин, 2-тио-зебуларин, 2-метоксицитидин, 2-метокси-5-метилцитидин, 4-метокси-псевдоизоцитидин, 4-метокси-1-метил-псевдоизоцитидин, 2-аминопурин, 2,6-диаминопурин, 7-деаза-аденин, 7-деаза-8-азааденин, 7-деаза-2-аминопурин, 7-деаза-8-аза-2-аминопурин, 7-деаза-2,6-диаминопурин, 7-деаза-8-аза-2,6-диаминопурин, 1-метиладенозин, N6-метиладенозин, N6-изопентениладенозин, N6- (цис-гидроксиизопентенил) аденозин, 2-метилтио-N6-(цис-гидроксиизопентенил) аденозин, N6-глицинилкарбамоиладенозин, N6-треонилкарбамоиладенозин, 2-метилтио-N6-треонилкарбамоиладенозин, N6,N6-диметиладенозин, 7-метиладенин, 2-метилтиоаденин, 2-метоксиаденин, инозин, 1-метилинозин, виозин, вибутозин, 7-деаза-гуанозин, 7-деаза-8-аза-гуанозин, 6-тио-гуанозин, 6-тио-7-деаза-гуанозин, 6-тио-7-деаза-8-азагуанозин, 7-метилгуанозин, 6-тио-7-метилгуанозин, 7-метилинозин, 6-метоксигуанозин, 1-метилгуанозин, N2-метилгуанозин, N2,N2-диметилгуанозин, 8-оксо-гуанозин, 7-метил-8-оксо-гуанозин, 1-метил-6-тио-гуанозин, N2-метил-6-тио-гуанозин и N2,N2-диметил-6-тио-гуанозин.
В одном варианте реализации мРНК содержит один или более модифицированных нуклеозидов, выбранных из группы, состоящей из следующих: псевдоуридин, пиридин-4-он-рибонуклеозид, 5-азауридин, 2-тио-5-азауридин, 2-тиоуридин, 4-тио-псевдоуридин, 2-тио-псевдоуридин, 5-гидроксиуридин, 3-метилуридин, 5 -карбоксиметил-уридин, 1-карбоксиметил-псевдоуридин, 5-пропинил-уридин, 1-пропинил-псевдоуридин, 5-тауринометилуридин, 1-тауринометил-псевдоуридин, 5-тауринометил-2-тио-уридин, 1-тауринометил-4-тиоуридин, 5-метил-уридин, 1-метил-псевдоуридин, 4-тио-1-метил-псевдоуридин, 2-тио-1-метил-псевдоуридин, 1-метил-1-деаза-псевдоуридин, 2-тио-1-метил-1-деаза-псевдоуридин, дигидроуридин, дигидропсевдоуридин, 2-тидигидроуридин, 2-тио-дигидропсевдуридин, 2-метоксиуридин, 2-метокси-4-тиоуридин, 4-метокси-псевдоуридин и 4-метокси 2-тио-псевдоуридин.
В одном варианте реализации мРНК содержит один или более модифицированных нуклеозидов, выбранных из группы, состоящей из следующих: 5-азацитидин, псевдоизоцитидин, 3-метилцитидин, N4-ацетилцитидин, 5-формилцитидин, N4-метилцитидин, 5-гидроксиметилцитидин, 1-метил-псевдоизоцитидин, пирроло-цитидин, пирроло-псевдоизоцитидин, 2-тиоцитидин, 2-тио-5-метилцитидин, 4-тио-псевдоизоцитидин, 4-тио-1-метил-псевдоизоцитидин, 4-тио-1-метил-1-деаза-псевдоизоцитидин, 1-метил-1-деаза-псевдоизоцитидин, зебуларин, 5-аза-зебуларин, 5-метилзебуларин, 5-аза-2-тио-зебуларин, 2-тио-зебуларин, 2-метоксицитидин, 2-метокси-5-метилцитидин, 4-метокси- псевдоизоцитидин и 4-метокси-1-метилпсевдоизоцитидин.
В одном варианте реализации мРНК содержит один или более модифицированных нуклеозидов, выбранных из группы, состоящей из следующих: 2-аминопурин, 2,6-диаминопурин, 7-деаза-аденин, 7-деаза-8-аза-аденин, 7-деаза-2-аминопурин, 7-деаза-8-аза-2-аминопурин, 7-деаза- 2,6-диаминопурин, 7-деаза-8-аза-2,6-диаминопурин, 1-метиладенозин, N6-метиладенозин, N6-изопентениладенозин, N6- (цис-гидроксиизопентенил) аденозин, 2-метилтио-N6-(цис- гидроксиизопентенил) аденозин, N6-глицинилкарбамоиладенозин, N6-треонилкарбамоиладенозин, 2-метилтио-N6-треонилкарбамоиладенозин, N6, N6-диметиладенозин, 7-метиладенин, 2-метилтиоаденин и 2-метоксиаденин.
В одном варианте реализации мРНК содержит один или более модифицированных нуклеозидов, выбранных из группы, состоящей из следующих: инозин, 1-метилинозин, виозин, вибутозин, 7-деаза-гуанозин, 7-деаза-8-аза-гуанозин, 6-тио-гуанозин, 6-тио-7-деаза-гуанозин, 6-тио-7- деаза-8-азагуанозин, 7-метилгуанозин, 6-тио-7-метилгуанозин, 7-метилинозин, 6-метоксигуанозин, 1-метилгуанозин, N2-метилгуанозин, N2, N2-диметилгуанозин, 8-оксогуанозин, 7-метил-8-оксогуанозин, 1-метил-6-тио-гуанозин, N2-метил-6-тио-гуанозин и N2, N2-диметил-6-тио-гуанозин.
В одном варианте реализации мРНК содержит один или более псевдоуридинов, один или более 5-метилцитозинов и/или один или более 5-метилцитидинов.
В одном варианте реализации мРНК содержит один или более псевдоуридинов.
В одном варианте реализации мРНК содержит один или более 5-метилцитидинов.
В одном варианте реализации мРНК содержит один или более 5-метилцитозинов.
В частных вариантах реализации мРНК, рассматриваемая в настоящей заявке, содержит поли(A)-хвост, помогающий защитить мРНК от деградации экзонуклеазой, стабилизировать мРНК и облегчить трансляцию. В некоторых вариантах реализации мРНК содержит 3' поли(А)-хвостовую структуру.
В частных вариантах реализации длина поли(А)-хвоста составляет по меньшей мере приблизительно 10, 25, 50, 75, 100, 150, 200, 250, 300, 350, 400, 450 или по меньшей мере приблизительно 500 или более адениновых нуклеотидов или любое промежуточное число адениновых нуклеотидов. В частных вариантах реализации длина поли(А)-хвоста составляет по меньшей мере приблизительно 125, 126, 127, 128, 129, 130, 131, 132, 133, 134, 135, 136, 137, 138, 139, 140, 141, 142, 143, 144, 145, 146, 147, 148, 149, 150, 151, 152, 153, 154, 155, 156, 157, 158, 159, 160, 161, 162, 163, 164, 165, 166, 167, 168, 169, 170, 171, 172, 173, 174, 175, 176, 177, 178, 179, 180, 181, 182, 183, 184, 185, 186, 187, 188, 189, 190, 191, 192, 193, 194, 195, 196, 197, 198, 199, 200, 201, 202, 202, 203, 205, 206, 207, 208, 209, 210, 211, 212, 213, 214, 215, 216, 217, 218, 219, 220, 221, 222, 223, 224, 225, 226, 227, 228, 229, 230, 231, 232, 233, 234, 235, 236, 237, 238, 239, 240, 241, 242, 243, 244, 245, 246, 247, 248, 249, 250, 251, 252, 253, 254, 255, 256, 257, 258, 259, 260, 261, 262, 263, 264, 265, 266, 267, 268, 269, 270, 271, 272, 273, 274 или 275 или более адениннуклеотидов.
В частных вариантах реализации длина поли(А)-хвоста составляет от приблизительно 10 до приблизительно 500 адениннуклеотидов, от приблизительно 50 до приблизительно 500 адениннуклеотидов, от приблизительно 100 до приблизительно 500 адениннуклеотидов, от приблизительно 150 до приблизительно 500 адениннуклеотидов, от приблизительно 200 до приблизительно 500 адениннуклеотидов, от приблизительно 250 до приблизительно 500 адениннуклеотидов, от приблизительно 300 до приблизительно 500 адениннуклеотидов, приблизительно 50 до приблизительно 450 адениннуклеотидов, от приблизительно 50 до приблизительно 400 адениннуклеотидов, от приблизительно 50 до приблизительно 350 адениннуклеотидов, от приблизительно 100 до приблизительно 500 адениннуклеотидов, от приблизительно 100 до приблизительно 450 адениннуклеотидов, от приблизительно 100 до приблизительно 400 адениннуклеотидов, от приблизительно 100 до приблизительно 350 адениннуклеотидов, от приблизительно 100 до приблизительно 300 адениннуклеотидов, от приблизительно 150 до приблизительно 500 адениннуклеотидов, от приблизительно 150 до приблизительно 450 адениннуклеотидов, от приблизительно 150 до приблизительно 400 адениннуклеотидов, от приблизительно 150 до приблизительно 350 адениннуклеотидов, от приблизительно 150 до приблизительно 300 адениннуклеотидов, от приблизительно 150 до приблизительно 250 адениннуклеотидов, от приблизительно 150 до приблизительно 200 адениннуклеотидов, от приблизительно 200 до приблизительно 500 адениннуклеотидов, от приблизительно 200 до приблизительно 450 адениннуклеотидов, от приблизительно 200 до приблизительно 400 адениннуклеотидов, от приблизительно 200 до приблизительно 350 адениннуклеотидов, от приблизительно 200 до приблизительно 300 адениннуклеотидов, от приблизительно 250 до приблизительно 500 адениннуклеотидов, от приблизительно 250 до приблизительно 450 адениннуклеотидов, от приблизительно 250 до приблизительно 400 адениннуклеотидов, от приблизительно 250 до приблизительно 350 адениннуклеотидов или от приблизительно 250 до приблизительно 300 адениннуклеотидов или любой промежуточный диапазон адениннуклеотидов.
Термины, которые описывают ориентацию полинуклеотидов, включают: «5'» (обычно конец полинуклеотида, имеющий свободную фосфатную группу) и «3'» (обычно конец полинуклеотида, имеющий свободную гидроксильную (OH) группу). Полинуклеотидные последовательности могут быть аннотированы в ориентации «от 5' до 3'» или «от 3' до 5'». Для ДНК и мРНК цепь от 5' до 3' обозначается как «смысловая», «плюс» или «кодирующая» цепь, поскольку ее последовательность идентична последовательности пре-мРНК [за исключением урацила (U) в РНК вместо тимина (T) в ДНК]. Для ДНК и мРНК комплементарную цепь от 3' до 5', которая представляет собой цепь, транскрибируемую РНК-полимеразой, обозначают как «матричная», «антисмысловая», «минус» или «некодирующая» цепь. Используемый в настоящей заявке термин «обратная ориентация» относится к последовательности от 5' до 3', записанной в ориентации от 3' до 5', или последовательности от 3' до 5', записанной в ориентации от 5' до 3'.
Термины «комплементарный» и «комплементарность» относятся к полинуклеотидам (то есть последовательности нуклеотидов), связанным правилами спаривания оснований. Например, комплементарной цепью для последовательности ДНК 5' A G T C A T G 3' будет 3' T C A G T A C 5'. Последняя последовательность часто пишется как обратный комплемент с 5'-концом слева и 3'-концом справа, 5' C A T G A C T 3'. Последовательность, которая идентична своему обратному комплементу, называется палиндромной последовательностью. Комплементарность может быть «частичной», при которой только некоторые основания нуклеиновых кислот сопоставляются в соответствии с правилами спаривания оснований. Либо между нуклеиновыми кислотами может быть «полная» комплементарность.
Используемый в настоящей заявке термин «кассета с нуклеиновой кислотой» или «кассета экспрессии» относится к генетическим последовательностям внутри вектора, которые могут экспрессировать РНК, а затем и полипептид. В одном варианте реализации кассета с нуклеиновой кислотой содержит представляющий интерес ген (-ы), например представляющий интерес полинуклеотид (-ы). В другом варианте реализации кассета с нуклеиновой кислотой содержит одну или более последовательностей контроля экспрессии, например, промотор, энхансер, поли(А) последовательность и представляющий интерес ген (-ы), например, представляющий интерес полинуклеотид (-ы). Векторы могут содержать 1, 2, 3, 4, 5, 6, 7, 8, 9 или 10 или более кассет с нуклеиновой кислотой. Кассета с нуклеиновой кислотой позиционно и последовательно ориентирована внутри вектора, так что нуклеиновая кислота в кассете может транскрибироваться в РНК и, при необходимости, транслироваться в белок или полипептид, подвергаться соответствующим посттрансляционным модификациям, необходимым для активности в трансформированной клетке и быть перемещенной в соответствующий компартмент для биологической активности путем нацеливания на соответствующие внутриклеточные компартменты или секреции во внеклеточные компартменты. Предпочтительно, кассета имеет 3'- и 5'-концы, адаптированные для готовой инсерции в вектор, например, она имеет сайты рестрикционной эндонуклеазы на каждом конце. В предпочтительном варианте реализации кассета с нуклеиновой кислотой содержит последовательность терапевтического гена, используемого для лечения, предотвращения или ослабления генетического нарушения. Кассета может быть удалена и вставлена в плазмиду или вирусный вектор как единое целое.
Полинуклеотиды включают представляющий интерес полинуклеотид (ы). Используемый в настоящей заявке термин «представляющий интерес полинуклеотид» относится к полинуклеотиду, кодирующему полипептид или слитый полипептид или полинуклеотид, который служит в качестве матрицы для транскрипции ингибирующего полинуклеотида, как это предусмотрено в настоящей заявке.
Кроме того, специалистам в данной области техники должно быть понятно, что в результате вырожденности генетического кода существует много нуклеотидных последовательностей, которые могут кодировать полипептид или его фрагмент варианта, как предполагается в настоящей заявке. Некоторые из этих полинуклеотидов обладают минимальной гомологией с нуклеотидной последовательностью любого нативного гена. Тем не менее, полинуклеотиды, которые варьируются вследствие различий в использовании кодонов, специально рассматриваются в частных вариантах реализации, например, полинуклеотиды, которые оптимизированы для селекции кодонов человека и/или приматов. В одном варианте реализации предусмотрены полинуклеотиды, содержащие специальные аллельные последовательности. Аллели представляют собой эндогенные полинуклеотидные последовательности, которые изменяются в результате одной или нескольких мутаций, таких как делеции, добавления и/или замены нуклеотидов.
В определенном варианте реализации представляющий интерес полинуклеотид содержит донорную матрицу репарации.
В определенном варианте реализации представляющий интерес полинуклеотид содержит ингибирующий полинуклеотид, включая, среди прочего, киРНК, миРНК, кшРНК, рибозим или другую ингибирующую РНК.
В одном варианте реализации донорная матрица репарации, содержащая ингибирующую РНК, содержит одну или более регуляторных последовательностей, таких как, например, сильный конститутивный pol III, например, промотор няРНК U6 человека или мыши, промотор H1 РНК человека и мыши или человеческий тРНК-val промотор или сильный конститутивный промотор pol II, как описано в настоящей заявке в другом месте.
Полинуклеотиды, рассматриваемые в частных вариантах реализации независимо от длины самой кодирующей последовательности, могут быть объединены с другими последовательностями ДНК, такими как промоторы и/или энхансеры, нетранслируемые области (UTR), последовательности Козака, сигналы полиаденилирования, дополнительные сайты ферментов рестрикции, множественные сайты клонирования, участки внутренней посадки рибосомы (IRES), сайты распознавания рекомбиназы (например, сайты LoxP, FRT и Att), терминирующие кодоны, сигналы терминации транскрипции, пост-транскрипционные элементы ответа и полинуклеотиды, кодирующие саморасщепляемый полипептид, метки эпитопа, описанные в другом месте в настоящей заявке или как известно в данной области техники, так что их общая длина может значительно варьироваться. Поэтому в частных вариантах реализации предполагается, что можно использовать полинуклеотидный фрагмент практически любой длины, причем общая длина предпочтительно ограничена простотой получения и использования в предполагаемом протоколе рекомбинантной ДНК.
Полинуклеотиды могут быть получены, обработаны, экспрессированы и/или доставлены с использованием любого из множества хорошо известных методов, известных и доступных в данной области. Чтобы экспрессировать желаемый полипептид, нуклеотидная последовательность, кодирующая полипептид, может быть вставлена в соответствующий вектор. Желаемый полипептид также может быть экспрессирован путем доставки в клетку мРНК, кодирующей данный полипептид.
Иллюстративные примеры векторов включают, среди прочего, плазмиду, автономно реплицируемые последовательности и транспонируемые элементы, например, Sleeping Beauty, PiggyBac.
Дополнительные иллюстративные примеры векторов включают, без ограничения, плазмиды, фагемиды, космиды, искусственные хромосомы, такие как дрожжевая искусственная хромосома (YAC), бактериальная искусственная хромосома (BAC) или P1-производная искусственная хромосома (PAC), бактериофаги, такие как фаг лямбда или фаг М13 и вирусы животных.
Иллюстративные примеры вирусов, полезных в качестве векторов, включают, без ограничения, ретровирус (включая лентивирус), аденовирус, адено-ассоциированный вирус, герпесвирус (например, вирус простого герпеса), поксвирус, бакуловирус, вирус папилломы и паповавирус (например, SV40).
Иллюстративные примеры векторов экспрессии включают, среди прочего, векторы pClneo (Promega) для экспрессии в клетках млекопитающих; pLenti4/V5-DEST™, pLenti6/V5-DEST™ и pLenti6.2/V5-GW/lacZ (Invitrogen) для лентивирус-опосредованного переноса и экспрессии генов в клетках млекопитающих. В частных вариантах реализации кодирующие последовательности полипептидов, раскрытые в настоящей заявке, могут быть лигированы в такие векторы экспрессии для экспрессии полипептидов в клетках млекопитающих.
В частных вариантах реализации вектор представляет собой эписомальный вектор или вектор, который поддерживается внехромосомно. Используемый в настоящей заявке термин «эписомальный» относится к вектору, который способен реплицироваться без интеграции в хромосомную ДНК хозяина и без постепенной потери из делящейся клетки-хозяина, а также означает, что указанный вектор реплицируется внехромосомно или эписомально.
«Последовательности контроля экспрессии», «элементы контроля» или «регуляторные последовательности», присутствующие в векторе экспрессии, представляют собой такие нетранслируемые области вектора: источник репликации, кассеты отбора, промоторы, энхансеры, интроны сигналов инициации трансляции (последовательность Shine Dalgarno или последовательность Козака), посттранскрипционные регуляторные элементы, последовательность полиаденилирования, 5'- и 3'-нетранслируемые области, которые взаимодействуют с белками клетки-хозяина для осуществления транскрипции и трансляции. Такие элементы могут различаться по своей силе и специфичности. В зависимости от используемой векторной системы и хозяина может использоваться любое количество подходящих элементов транскрипции и трансляции, включая убиквитарные промоторы и индуцибельные промоторы.
В частных вариантах реализации полинуклеотид содержит вектор, включая, среди прочего, векторы экспрессии и вирусные векторы. Вектор может содержать одну или более экзогенных, эндогенных или гетерологичных контрольных последовательностей, таких как промоторы и/или энхансеры. «Эндогенная контрольная последовательность» представляет собой последовательность, которая естественным образом связана с данным геном в геноме. «Экзогенная контрольная последовательность» представляет собой последовательность, которая помещается в сопоставление с геном посредством генетических манипуляций (то есть молекулярно-биологических методов), так что транскрипция указанного гена направляется связанным энхансером/промотором. «Гетерологичная контрольная последовательность» представляет собой экзогенную последовательность, которая принадлежит к другому виду, нежели генетически манипулируемая клетка. «Синтетическая» контрольная последовательность может содержать элементы еще одной эндогенной и/или экзогенной последовательности и/или последовательности, определенные in vitro или in silico, которые обеспечивают оптимальную промоторную и/или энхансерную активность для конкретной терапии.
Используемый в настоящей заявке термин «промотор» относится к сайту распознавания полинуклеотида (ДНК или РНК), с которым связывается РНК-полимераза. РНК-полимераза инициирует и транскрибирует полинуклеотиды, функционально связанные с промотором. В частных вариантах реализации промоторы, функциональные в клетках млекопитающих, содержат AT-богатую область, расположенную на расстоянии приблизительно от 25 до 30 оснований в направлении против хода транскрипции от сайта, где инициируется транскрипция, и/или другую последовательность, расположенную на расстоянии от 70 до 80 оснований в направлении против хода транскрипции от начала транскрипции, область CNCAAT, где N может быть любым нуклеотидом.
Термин «энхансер» относится к сегменту ДНК, который содержит последовательности, способные обеспечивать усиленную транскрипцию, и в некоторых случаях способные функционировать независимо от их ориентации относительно другой контрольной последовательности. Энхансер может функционировать совместно или аддитивно с промоторами и/или другими энхансерными элементами. Термин «промотор/энхансер» относится к сегменту ДНК, который содержит последовательности, способные обеспечивать функции как промотора, так и энхансера.
Термин «функционально связанный» относится к сопоставлению, в котором описанные компоненты находятся в взаимосвязи, позволяющей им функционировать по своему назначению. В одном варианте реализации этот термин относится к функциональной связи между последовательностью контроля экспрессии нуклеиновой кислоты (такой как промотор и/или энхансер) и второй полинуклеотидной последовательностью, например, представляющим интерес полинуклеотидом, когда последовательность контроля экспрессии направляет транскрипцию экспрессии нуклеиновой кислоты, соответствующей второй последовательности.
Используемый в настоящей заявке термин «последовательность контроля конститутивной экспрессии» относится к промотору, энхансеру или промотору/энхансеру, который длительно или непрерывно обеспечивает транскрипцию функционально связанной последовательности. Конститутивная последовательность контроля экспрессии может быть «убиквитарным» промотором, энхансером или промотором/энхансером, который обеспечивает экспрессию в широком разнообразии типов клеток и тканей, или «специфичным для клетки», «специфичным для типа клеток», «специфичным для клеточных линий» или «тканеспецифичным» промотором, э энхансером или промотором/энхансером, который обеспечивает экспрессию в ограниченном разнообразии типов клеток и тканей, соответственно.
Иллюстративные убиквитарные последовательности контроля экспрессии, подходящие для использования в частных вариантах реализации, включают, среди прочего, немедленно-ранний промотор цитомегаловируса (CMV), промотор обезьяньего вируса 40 (SV40) (например, ранний или поздний), LTR промотор вирус мышиного лейкоза Молони (MoMLV), LTR промотор вируса саркомы Рауса (RSV), промотор вируса простого герпеса (HSV) (тимидинкиназа), промоторы H5, P7.5 и P11 из вируса осповакцины, промотор короткого фактора элонгации 1-альфа (EF1a- short), промотор длинного фактора элонгации 1-альфа (EF1a-long), ранний ответ роста 1 (EGR1), ферритин H (FerH), ферритин L (FerL), глицеральдегид-3-фосфатдегидрогеназа (GAPDH), эукариотический фактор инициации трансляции 4A1 (EIF4A1), белок теплового шока 5 70 кДа (HSPA5), белок теплового шока бета 90 кДа, член 1 (HSP90B1), белок теплового шока 70 кДа (HSP70), β-кинезин (β-KIN), человеческий локус ROSA 26к (Irions et al., Nature Biotechnology 25, 1477 - 1482 (2007)), промотор убиквитина С (UBC), промотор фосфоглицерат киназы-1 (PGK), промотор энхансера цитомегаловируса/β-актина курицы (CAG), промотор β-актина и промотор энхансера вируса миелопролиферативной саркомы, с удаленным участок отрицательного контроля, с замещенным сайтом связывания праймера dl587rev (MND) (Challita et al., J Virol. 69(2):748-55 (1995)).
В частном варианте реализации может быть желательным использовать последовательность контроля экспрессии, специфичную к клетке, типу клетки, клеточной линии или ткани, для достижения специфической для типа клеток, специфичной к линии или тканевой экспрессии желаемой полинуклеотидной последовательности (например, для экспрессии конкретной нуклеиновой кислоты, кодирующей полипептид только в определенном подмножестве типов клеток, клеточных линий или тканей или на определенных стадиях развития).
«Условная экспрессия» в настоящей заявке может относиться к любому типу условной экспрессии, включая, среди прочего, индуцибельную экспрессию; репрессируемую экспрессию; экспрессию в клетках или тканях, имеющих конкретное физиологическое, биологическое или болезненное состояние и т.д. Это определение не предназначено для исключения специфической к типу клеток или тканей экспрессии. Некоторые варианты реализации предусматривают условную экспрессию рассматриваемого полинуклеотида, например, экспрессия контролируется путем воздействия на клетку, ткань, организм и т.д. обработки или состояния, которое вызывает экспрессию полинуклеотида или которое вызывает увеличение или уменьшение экспрессии полинуклеотида, кодируемого рассматриваемым полинуклеотидом.
Иллюстративные примеры индуцибельных промоторов/систем включают, среди прочего, стероид-индуцибельные промоторы, такие как промоторы для генов, кодирующих глюкокортикоидные или эстрогенные рецепторы (индуцируемые обработкой соответствующим гормоном), металлотиониновый промотор (индуцируемый обработкой различными тяжелыми металлами), промотор MX-1 (индуцируемый интерфероном), мифепристон-регулируемая система GeneSwitch (Sirin et al., 2003, Gene, 323:67), индуцируемый куматом генный переключатель (WO 2002/088346), тетрациклин-зависимые регуляторные системы и т.д.
Условная экспрессия также может быть достигнута с использованием сайт-специфической ДНК-рекомбиназы. Согласно некоторым вариантам реализации полинуклеотиды содержат по меньшей мере один (обычно два) сайт (сайта) для рекомбинации, опосредованной сайт-специфической рекомбиназой. Используемые в настоящей заявке термины «рекомбиназа» или «сайт-специфическая рекомбиназа» включают в себя эксцизивные или интегративные белки, ферменты, кофакторы или ассоциированные белки, которые участвуют в реакциях рекомбинации с участием одного или нескольких сайтов рекомбинации (например, двух, трех, четырех, пяти, шести, семи, восьми, девяти, десяти или более), которые могут быть белками дикого типа (см. Landy, Current Opinion in Biotechnology 3: 699-707 (1993)) или мутантами, производными (например, слитые белки, содержащие последовательности рекомбинационных белков или их фрагменты), фрагменты и их варианты. Иллюстративные примеры рекомбиназ, подходящих для использования В частных вариантах реализации включают, среди прочего: Cre, Int, IHF, Xis, Flp, Fis, Hin, Gin, C31, Cin, Tn3 resolvase, TndX, XerC, XerD, TnpX, Hjc, Gin, SpCCE1 и ParA.
Полинуклеотиды могут содержать один или более сайтов рекомбинации для любой из множества сайт-специфических рекомбиназ. Следует понимать, что сайт-мишень для сайт-специфической рекомбиназы является дополнением к любому сайту (-ам), необходимому для интеграции вектора, например, ретровирусного вектора или лентивирусного вектора. Используемые в настоящей заявке термины «последовательность рекомбинации», «сайт рекомбинации» или «сайт сайт-специфической рекомбинации» относятся к конкретной нуклеотидной последовательности, которую рекомбиназа распознает и с которой связывается.
Например, один сайт рекомбинации для Cre-рекомбиназы представляет собой loxP, который представляет собой последовательность из 34 пар оснований, содержащую два инвертированных повтора из 13 пар оснований (служащих сайтами связывания рекомбиназы), фланкирующих сердцевинную последовательность из 8 пар оснований (см. Фиг.1 из Sauer, B., Current Opinion in Biotechnology 5:521-527 (1994)). Другие иллюстративные сайты loxP включают, среди прочего: lox511 (Hoess et al., 1996; Bethke and Sauer, 1997), lox5171 (Lee and Saito, 1998), lox2272 (Lee and Saito, 1998), m2 (Langer et al., 2002), lox71 (Albert et al., 1995) и lox66 (Albert et al., 1995).
Подходящие сайты распознавания для рекомбиназы FLP включают, среди прочего: FRT (McLeod, et al., 1996), F1, F2, F3 (Schlake and Bode, 1994), F4, F5 (Schlake and Bode, 1994), FRT(LE) (Senecoff et al., 1988), FRT(RE) (Senecoff et al., 1988).
Другими примерами последовательностей распознавания являются последовательности attB, attP, attL и attR, которые распознаются рекомбиназным ферментом λ-интеграза, например, phi-c31. SSR φC31 обеспечивает рекомбинацию только между гетеротипическими сайтами attB (длиной 34 п.н.) и attP (длиной 39 п.н.) (Groth et al., 2000). attB и attP, названные по сайтам присоединения для фаговой интегразы на бактериальном и фаговом геномах соответственно, оба содержат несовершенные инвертированные повторы, которые, вероятно, связаны гомодимерами φC31 (Groth et al., 2000). Сайты продуктов, attL и attR, эффективно инертны к дальнейшей φC31-опосредованной рекомбинации (Belteki et al., 2003), что делает реакцию необратимой. Было обнаружено, что для катализирования инсерции ДНК, несущая attB, встраивается в геномный сайт attP легче, чем сайт attP в геномный сайт attB (Thyagarajan et al., 2001; Belteki et al., 2003). Таким образом, типичные стратегии помещают путем гомологичной рекомбинации «стыковочный сайт», несущий attP, в определенный локус, который затем соединяется с входящей последовательностью, несущей attB, для инсерции.
В одном варианте реализации рассматриваемый в настоящей заявке полинуклеотид содержит донорную матрицу репарации полинуклеотида, фланкированную парой сайтов распознавания рекомбиназы. в частных вариантах реализации, репарационный матричный полинуклеотид фланкирован сайтами LoxP, сайтами FRT или сайтами att.
В частных вариантах реализации полинуклеотиды, рассматриваемые в настоящей заявке, включают один или более представляющих интерес полинуклеотидов, которые кодируют один или более полипептидов. В частных вариантах, чтобы добиться эффективной трансляции каждого из множества полипептидов, полинуклеотидные последовательности могут быть разделены одной или несколькими последовательностями IRES или полинуклеотидными последовательностями, кодирующими саморасщепляемый полипептид.
Используемый в настоящей заявке термин «участок внутренней посадки рибосомы» или «IRES» относится к элементу, который способствует прямому внутреннему входу рибосомы в инициирующий кодон, такой как ATG, цистрона (области, кодирующей белок), тем самым приводя к кап-независимой трансляции гена. См., например, Jackson et al., 1990. Trends Biochem Sci 15 (12): 477-83) и Jackson and Kaminski. 1995. RNA 1 (10): 985-1000. Примеры IRES, обычно используемых специалистами в данной области, включают в себя описанные в патенте США №6 692 736. Дополнительные примеры «IRES», известные в данной области, включают, среди прочего, IRES, получаемый из пикорнавируса (Jackson et al., 1990), и IRES, получаемый из вирусных или клеточных источников мРНК, таких как, например, белок, связывающий тяжелую цепь иммуноглобулина (BiP), фактор роста эндотелия сосудов (VEGF) (Huez et al. 1998. Mol. Cell. Biol. 18 (11): 6178-6190), фактор роста фибробластов 2 (FGF-2) и инсулиноподобный фактор роста (IGFII), фактор инициации трансляции eIF4G и дрожжевые транскрипционные факторы TFIID и HAP4, вирус энцефеломикардита (EMCV), поставляемый на рынок Novagen (Duke et al., 1992. J. Virol 66 (3): 1602-9) и VEGF IRES (Huez et al., 1998. Mol Cell Biol 18 (11): 6178-90). Также сообщалось об IRES в вирусных геномах видов Picornaviridae, Dicistroviridae и Flaviviridae и в HCV, вирусе мышиного лейкоза Friend (FrMLV) и вирусе мышиного лейкоза Moloney (MoMLV).
В одном варианте реализации IRES, используемый в полинуклеотидах, рассматриваемых в настоящей заявке, представляет собой EMCV IRES.
В частных вариантах реализации полинуклеотиды содержат полинуклеотиды, имеющие консенсусную последовательность Козака и кодирующие желаемый полипептид. Используемый в настоящей заявке термин «последовательность Козака» относится к короткой нуклеотидной последовательности, которая значительно облегчает начальное связывание мРНК с небольшой субъединицей рибосомы и увеличивает трансляцию. Консенсусная последовательность Козака представляет собой (GCC) RCCATGG (SEQ ID NO: 105), где R представляет собой пурин (A или G) (Kozak, 1986. Cell. 44(2):283-92 и Kozak, 1987. Nucleic Acids Res. 15(20):8125-48).
Элементы, направляющие эффективную терминацию и полиаденилирование гетерологичных транскриптов нуклеиновых кислот, увеличивают экспрессию гетерологичных генов. Сигналы терминации транскрипции обычно находятся в прямом направлении от сигнала полиаденилирования. В частных вариантах реализации векторы содержат последовательность полиаденилирования 3 ' полинуклеотида, кодирующего полипептид, подлежащий экспрессии. Используемые в настоящей заявке термины «сайт polyA», «последовательность polyA», «сайт poly(A)» или «последовательность poly(A)» означают последовательность ДНК, которая направляет как терминацию, так и полиаденилирование растущего РНК-транскрипта с помощью РНК-полимеразы II. Последовательности полиаденилирования могут способствовать стабильности мРНК путем добавления поли (А)-хвоста к 3'-концу кодирующей последовательности и, таким образом, способствовать повышению эффективности трансляции. Эффективное полиаденилирование рекомбинантного транскрипта является желательным, поскольку транскрипты, лишенные поли(А)-хвоста, нестабильны и быстро деградируют.Иллюстративные примеры поли(А)-сигналов, которые можно использовать в векторе, включают идеальную поли(А) -последовательность (например, AATAAA, ATTAAA, AGTAAA), бычью поли(А)-последовательность гормона роста (BGHpA), кроличью β -глобин-поли(А)-последовательность (rβgpA) или другую подходящую гетерологичную или эндогенную поли(А)-последовательность, известную в данной области техники.
В некоторых вариантах реализации полинуклеотид или клетка, несущая полинуклеотид, использует ген самоубийства, включая индуцибельный ген самоубийства, для снижения риска прямой токсичности и/или неконтролируемой пролиферации. В частных вариантах реализации ген самоубийства не является иммуногенным для хозяина, несущего указанный полинуклеотид или клетку. Определенным примером гена самоубийства, который может быть использован, является каспаза-9 или каспаза-8 или цитозин-деаминаза. Каспазу-9 можно активировать с помощью специального химического индуктора димеризации (CID).
В некоторых аспектах полинуклеотиды содержат генные сегменты, которые делают генетически модифицированные клетки, рассматриваемые в настоящей заявке, восприимчивыми к отрицательному отбору in vivo. Термин «отрицательный отбор» относится к инфузированной клетке, которая может быть элиминирована в результате изменения in vivo-состояния индивидуума. Отрицательный селектируемый фенотип может быть результатом инсерции гена, который придает чувствительность вводимому агенту, например, соединению. Гены отрицательной селекции известны в данной области и включают, среди прочего: ген тимидинкиназы вируса простого герпеса типа I (HSV-I TK), который придает чувствительность к ганцикловиру; ген клеточной гипоксантинфосфрибозилтрансферазы (HPRT), ген клеточной аденинфосфорибозилтрансферазы (APRT) и ген бактериальной цитозин-деаминазы.
В некоторых вариантах реализации генетически модифицированные клетки содержат полинуклеотид, дополнительно содержащий положительный маркер, который делает возможным отбор клеток отрицательного селектируемого фенотипа in vitro. Положительный селектируемый маркер может представлять собой ген, который после введения в клетку-хозяина экспрессирует доминантный фенотип, позволяющий осуществлять положительный отбор клеток, несущих указанный ген. Гены этого типа известны в данной области и включают, среди прочего, ген гигромицин-B-фосфотрансферазы (hph), который придает устойчивость к гигромицину B, ген аминогликозид-фосфотрансферазы (neo или aph) из Tn5, который кодирует устойчивость к антибиотику G418, ген дигидрофолатредуктазы (DHFR), ген аденозиндеаминазы (ADA) и ген множественной лекарственной устойчивости (MDR).
В одном варианте реализации положительный селектируемый маркер и отрицательный селектируемый элемент связаны так, что потеря отрицательного селектируемого элемента обязательно также сопровождается потерей положительного селектируемого маркера. В частном варианте реализации положительные и отрицательные селектируемые маркеры слиты таким образом, что потеря одного обязательно ведет к потере другого. Примером слитого полинуклеотида, который дает в качестве продукта экспрессии полипептид, который обладает желаемыми положительными и отрицательными признаками отбора, описанными выше, является гибридный ген гигромицин-фосфотрансферазы тимидинкиназы (HyTK). Экспрессия указанного гена дает полипептид, который придает устойчивость к гигромицину B для положительного отбора in vitro и чувствительность к ганцикловиру для отрицательного отбора in vivo. См. также публикации PCT US91/08442 и PCT/US94/05601 автора S.D. Lupton, описывающие использование бифункциональных селектируемых слитых генов, полученных в результате слияния доминантных позитивных селектируемых маркеров с отрицательными селектируемыми маркерами.
Предпочтительные положительные селектируемыые маркеры происходят из генов, выбранных из группы, состоящей из hph, nco и gpt, а предпочтительные отрицательные селектируемыые маркеры происходят из генов, выбранных из группы, состоящей из цитозин-деаминазы, HSV-I TK, VZV TK, HPRT, APRT и gpt. Типичные бифункциональные селектируемые слитые гены, рассматриваемые в частных вариантах реализации включают, среди прочего, гены, в которых положительный селектируемый маркер происходит из hph или neo, а отрицательный селектируемый маркер происходит из цитозин-деаминазы или гена TK или селектируемого маркера.
В частных вариантах реализации полинуклеотиды, кодирующие один или более вариантов нуклеаз, megaTAL, ферментов, изменяющих структуру концов, или слитые полипептиды, могут быть введены в гемопоэтические клетки, например Т-клетки, как невирусными, так и вирусными способами. В частных вариантах реализации доставка одного или нескольких полинуклеотидов, кодирующих нуклеазы, и/или донорной матрицы репарации, может обеспечиваться одним и тем же способом или разными способами, и/или одним и тем же вектором или разными векторами.
Термин «вектор» используется в настоящей заявке для обозначения молекулы нуклеиновой кислоты, способной переносить или транспортировать другую молекулу нуклеиновой кислоты. Перенесенная нуклеиновая кислота, как правило, связана с молекулой вектора нуклеиновой кислоты, например, встроена в нее. Вектор может включать последовательности, которые управляют автономной репликацией в клетке, или может включать последовательности, достаточные для интеграции в ДНК клетки-хозяина. В частных вариантах реализации для доставки одного или нескольких полинуклеотидов, рассматриваемых в настоящей заявке, в Т-клетку, используются невирусные векторы.
Иллюстративные примеры невирусных векторов включают, среди прочего, плазмиды (например, ДНК-плазмиды или РНК-плазмиды), транспозоны, космиды и бактериальные искусственные хромосомы.
Иллюстративные способы невирусной доставки полинуклеотидов, рассматриваемые в частных вариантах реализации включают, среди прочего: электропорацию, сонопорацию, липофекцию, микроинъекцию, биолистику, виросомы, липосомы, иммунолипосомы, наночастицы, поликатион или конъюгаты липид:нуклеиновая кислота, «оголенную» ДНК, искусственные вирионы, DEAE-декстран-опосредованный перенос, генную пушку и тепловой шок.
Иллюстративные примеры систем доставки полинуклеотидов, подходящих для использования в частных вариантах реализации, рассматриваемых в настоящей заявке, включают, среди прочего, предоставленные Amaxa Biosystems, Maxcyte, Inc., BTX Molecular Delivery Systems, ThermoFisher Scientific и Copernicus Therapeutics Inc. Реагенты для липофекции имеются на рынке (например, Transfectam™ и Lipofectin™). Катионные и нейтральные липиды, которые подходят для эффективной липофекции полинуклеотидов с распознаванием рецепторов, описаны в литературе. См например, Liu et al. (2003) Gene Therapy. 10:180-187; и Balazs et al. (2011) Journal of Drug Delivery. 2011:1-12. В частных вариантах реализации также предусмотрена направленная на антитела доставка на основе неживых наноклеток, полученных из бактерий.
Вирусные векторы, содержащие полинуклеотиды, рассматриваемые в частных вариантах реализации могут доставляться in vivo путем введения отдельному пациенту, обычно путем системного введения (например, внутривенной, внутрибрюшинной, внутримышечной, подкожной или внутричерепной инфузии) или местного применения, как описано ниже. В альтернативном варианте векторы могут доставляться в клетки ex vivo, например в клетки, эксплантированные от отдельного пациента (например, мобилизованная периферическая кровь, лимфоциты, аспираты костного мозга, биопсия ткани и т.д.), или универсальные донорские гематопоэтические стволовые клетки с последующей реимплантацией клетки в пациента.
В одном варианте реализации вирусные векторы, содержащие варианты нуклеазы и/или донорную матрицу репарации, вводят непосредственно в организм для трансдукции клеток in vivo. В альтернативном варианте можно вводить оголенную ДНК. Введение осуществляют любым путем, обычно используемым для введения молекулы в окончательный контакт с клетками крови или ткани, включая, без ограничения, инъекцию, инфузию, местное применение и электропорацию. Подходящие способы введения таких нуклеиновых кислот доступны и хорошо известны специалистам в данной области, и, хотя для введения конкретной композиции можно использовать более одного пути, конкретный путь часто может обеспечить более непосредственную и более эффективную реакцию, чем другой путь.
Иллюстративные примеры вирусных векторных систем, подходящих для использования в частных вариантах реализации, рассматриваемых в настоящей заявке, включают, среди прочего, векторы адено-ассоциированного вируса (AAV), ретровирус, вируса простого герпеса, аденовируса и вируса осповакцины.
В различных вариантах реализации один или более полинуклеотидов, кодирующих вариант нуклеазы и/или донорную матрицу репарации, вводят в гематопоэтическую клетку, например, Т-клетку, путем трансдукции клетки рекомбинантным аденоассоциированным вирусом (rAAV), содержащим один или больше полинуклеотидов.
AAV представляет собой небольшой (~ 26 нм) дефектный по репликации вирус, в основном эписомальный, без оболочки. AAV может инфицировать как делящиеся, так и неделящиеся клетки и может встраивать свой геном в геном клетки-хозяина. Рекомбинантные AAV (rAAV) обычно состоят, как минимум, из трансгена и его регуляторных последовательностей, а также 5'- и 3'-инвертированных концевых повторов AAV (ITR). Последовательности ITR имеют длину приблизительно 145 п.н. Векторы rAAV, содержащие два ITR, имеют полезную нагрузку приблизительно 4,4 т.п.н.
Самокомплементарные векторы rAAV содержат третью ITR и упаковывают две цепи рекомбинантной части вектора, оставляя только приблизительно 2,1 т.п.н для рассматриваемых в настоящей заявке полинуклеотидов. В одном варианте реализации вектор AAV представляет собой вектор scAAV.
Расширенные возможности упаковки, которые приблизительно вдвое превышают емкость упаковки rAAV (приблизительно 9 т.п.н), были достигнуты с использованием двойных векторных стратегий rAAV. Двойные векторные стратегии, полезные для получения rAAV, рассматриваемые в настоящей заявке, включают, среди прочего, сплайсинг (транс-сплайсинг), гомологичную рекомбинацию (перекрытие) или комбинацию этих двух стратегий (гибридная). В стратегии двойного AAV-транс-сплайсинга донорный сигнал сплайсинга (SD) размещают на 3'-конце 5'-половинного вектора, а акцепторный сигнал (SA) сплайсинга размещают на 5'-конце 3'- половинного вектора. При коинфицировании одной и той же клетки двойными AAV векторами и конкатемеризацией «голова к хвосту» двух половин, опосредованной инвертированным концевым повторением (ITR), транс-сплайсинг приводит к образованию зрелой мРНК и полноразмерного белка (Yan et al, 2000). Трансплайсинг успешно используется для экспрессии крупных генов в мышцах и сетчатке (Reich et al, 2003; Lai et al, 2005). В альтернативном варианте две половины большой трансгенной кассеты экспрессии, содержащейся в двойных векторах AAV, могут содержать гомологичные перекрывающиеся последовательности (на 3'-конце 5'-половинного вектора и на 5'-конце 3'-половинного вектора, перекрытие двойных AAV), что будет способствовать восстановлению одного большого генома путем гомологичной рекомбинации (Duan et al, 2001). Эта стратегия зависит от рекомбиногенных свойств трансгенных перекрывающихся последовательностей (Ghosh et al, 2006). Третья стратегия двойных AAV (гибридная) основана на добавлении высоко рекомбиногенной области от экзогенного гена (то есть, щелочной фосфатазы; Ghosh et al., 2008, Ghosh et al, 2011)) к векторам транс-сплайсинга. Указанная добавленная область располагается в прямом направлении от сигнала SD в 5'-половинном векторе и в направлении против хода транскрипции сигнала S A в 3'-половинном векторе для увеличения рекомбинации между двойными AAV.
Термины «гибридный AAV», «гибридный rAAV», «химерный AAV» или «химерный rAAV» относятся к геному rAAV, упакованному с капсидом другого серотипа AAV (и предпочтительно, другого серотипа из одного или нескольких ITR AAV), который в других случаях может упоминаться как псевдотипизированный rAAV. Например, геном rAAV типа 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15 или 16 может быть инкапсидирован в капсид AAV типа 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15 или 16 или его вариант, при условии, что капсид AAV и геном (и, предпочтительно, один или более ITR AAV)) имеют разные серотипы. В некоторых случаях можно псевдотипированную частицу rAAV можно обозначить как типа «x/y», где «x» указывает источник ITR, а «y» указывает серотип капсида, например, 2/5 rAAV. частица имеет ITR от AAV2 и капсид от AAV6.
В частных вариантах реализации rAAV содержит ITR и капсидные последовательности, выделенные из AAV1, AAV2, AAV3, AAV4, AAV5, AAV6, AAV7, AAV8, AAV9, AAV10, AAV11, AAV 12, AAV13, AAV 14, AAV15 и AAV16.
В некоторых вариантах реализации используется химерный rAAV, где последовательности ITR выделены из одного серотипа AAV, а последовательности капсида выделены из другого серотипа AAV. Например, rAAV с последовательностями ITR, полученными из AAV2, и капсидными последовательностями, полученными из AAV6, называют AAV2/AAV6. В частных вариантах реализации вектор rAAV может содержать ITR из AAV2 и капсидные белки из любого из AAV1, AAV2, AAV3, AAV4, AAV5, AAV6, AAV7, AAV8, AAV9 или AAV10. В предпочтительном варианте реализации rAAV содержит последовательности ITR, полученные из AAV2, и капсидные последовательности, полученные из AAV6. В предпочтительном варианте реализации rAAV содержит последовательности ITR, полученные из AAV2, и капсидные последовательности, полученные из AAV2.
В некоторых вариантах реализации к капсидам AAV могут быть применены сконструированные и селекционные методы, чтобы сделать их более способными к трансдукции изучаемых клеток.
Конструирование векторов rAAV, их получение и очистка описаны, например, в патентах США №№9169494; 9169492; 9012224; 8889641; 8809058; и 8784999, каждый из которых включен в настоящее описание в качестве ссылки в полном объеме.
В различных вариантах реализации один или более полинуклеотидов, кодирующих вариант нуклеазы и/или донорную матрицу репарации, вводят в гемопоэтическую клетку путем трансдукции клетки ретровирусом, например, лентивирусом, содержащим один или более полинуклеотидов.
Используемый в настоящей заявке термин «ретровирус» относится к РНК-вирусу, который осуществляет обратную транскрипцию своей геномной РНК в линейную двухцепочечную копию ДНК и впоследствии ковалентно интегрирует свою геномную ДНК в геном хозяина. Иллюстративные ретровирусы, подходящие для использования в частных вариантах реализации, включают, среди прочего: вирус мышиного лейкоза Молони (M-MuLV), вирус мышиной саркомы Молони (MoMSV), вирус мышиной саркомы Харви (HaMuSV), вирус опухоли молочной железы мыши (MuMTV), вирус лейкоза гиббонов (GaLV), вирус лейкоза кошек (FLV), спумавирус, вирус лейкоза мышей Френда, вирус мышиных стволовых клеток (MSCV) и вирус саркомы Рауса (RSV) и лентивирус.
Используемый в настоящей заявке термин «лентивирус» относится к группе (или роду) сложных ретровирусов. Иллюстративные лентивирусы включают, среди прочего: ВИЧ (вирус иммунодефицита человека; включая ВИЧ типа 1 и ВИЧ типа 2); вирус висна-маеди (VMV); вирус артрита-энцефалита коз и овец (CAEV); вирус инфекционной анемии лошадей (EIAV); вирус иммунодефицита кошек (FIV); вирус иммунодефицита крупного рогатого скота (BIV); и вирус иммунодефицита обезьян (SIV). В одном варианте реализации являются предпочтительными векторные каркасные цепи на основе ВИЧ (то есть элементы цис-действующей последовательности ВИЧ).
В различных вариантах реализации рассматриваемый в настоящей заявке лентивирусный вектор содержит один или более LTR и один или более или все из следующих вспомогательных элементов: cPPT/FLAP, сигнал упаковки Psi (Ψ), элемент экспорта, poly(A)-последовательности, и могут, необязательно, содержать WPRE или HPRE, инсуляторный элемент, селектируемый маркер и ген самоубийства клетки, как обсуждается в другом месте настоящей заявки.
В частных вариантах реализации лентивирусные векторы, рассматриваемые в настоящей заявке, могут быть интегрирующим или неинтегрирующим или дефектным по интеграции лентивирусом. Используемый в настоящей заявке термин «дефектный по интеграции лентивирус» или «IDLV» относится к лентивирусу, имеющему интегразу, которая не обладает способностью интегрировать вирусный геном в геном клеток-хозяев. Некомпетентные по интеграции вирусные векторы были описаны в заявке на патент WO 2006/010834, которая полностью включена в настоящее описание посредством ссылки.
Иллюстративные мутации в гене pol ВИЧ-1, подходящие для снижения активности интегразы, включают, среди прочего: H12N, H12C, H16C, H16V, S81 R, D41A, K42A, H51A, Q53C, D55V, D64E, D64V, E69A, K71A, E85A, E87A, D116N, D1161, D116A, N120G, N1201, N120E, E152G, E152A, D35E, K156E, K156A, E157A, K159E, K159A, K160A, R166A, D167A, E170A, H171A, K173A, K186Q, K186T, K188T, E198A, R199c, R199T, R199A, D202A, K211A, Q214L, Q216L, Q221 L, W235F, W235E, K236S, K236A, K246A, G247W, D253A, R262A, R263A и K264H.
В одном варианте реализации ген pol с дефицитом интегразы ВИЧ-1 содержит мутацию D64V, D116I, D116A, E152G или E152A; мутации D64V, D116I и E152G; или мутации D64V, D116A и E152A.
В одном варианте реализации ген pol с дефицитом интегразы ВИЧ-1 содержит мутацию D64V.
Термин «длинный концевой повтор (LTR)» относится к доменам пар оснований, расположенным на концах ретровирусных ДНК, которые в контексте своей естественной последовательности являются прямыми повторами и содержат области U3, R и U5.
Используемый в настоящей заявке термин «элемент FLAP» или «cPPT/FLAP» относится к нуклеиновой кислоте, последовательность которой включает в себя центральный полипуриновый тракт и центральные концевые последовательности (cPPT и CTS) ретровируса, например, ВИЧ-1 или ВИЧ-2. Подходящие элементы FLAP описаны в патенте США №6 682 907 и в Zennou et al., 2000, Cell, 101: 173. В другом варианте реализации лентивирусный вектор содержит элемент FLAP с одной или несколькими мутациями в элементах cPPT и/или CTS. В еще одном варианте реализации лентивирусный вектор содержит либо cPPT, либо CTS элемент.В еще одном варианте реализации лентивирусный вектор не содержит cPPT или CTS элемент.
Используемый в настоящей заявке термин «сигнал упаковки» или «последовательность упаковки» относится к последовательностям psi [Ψ], расположенным в ретровирусном геноме, которые необходимы для инсерции вирусной РНК в вирусный капсид или частицу, см., например,, Clever et al., 1995. J. of Virology, Vol.69, No. 4; pp.2101-2109.
Термин «экспортный элемент» относится к цис-действующему посттранскрипционному регуляторному элементу, который регулирует транспорт РНК-транскрипта из ядра в цитоплазму клетки. Примеры элементов экспорта РНК включают, среди прочего, элемент ответа rev (RRE) вируса иммунодефицита человека (ВИЧ) (см., например, Cullen et al., 1991. J. Virol. 65: 1053; и Cullen et al., 1991. Cell 58: 423), и посттранскрипционный регуляторный элемент вируса гепатита В (HPRE).
В частных вариантах реализации экспрессию гетерологичных последовательностей в вирусных векторах увеличивается путем встраивания посттранскрипционных регуляторных элементов, эффективных сайтов полиаденилирования и, необязательно, сигналов терминации транскрипции в векторы. Разнообразные посттранскрипционные регуляторные элементы могут повышать экспрессию гетерологичной нуклеиновой кислоты в белке, например, посттранскрипционный регуляторный элемент вируса гепатита сурка (WPRE; Zufferey et al., 1999, J. Virol., 73:2886); посттранскрипционный регуляторный элемент, присутствующий в вирусе гепатита В (HPRE) (Huang et al., Mol. Cell. Biol., 5:3864); и тому подобное (Liu et al., 1995, Genes Dev., 9:1766).
Лентивирусные векторы предпочтительно содержат несколько усовершенствований в отношении безопасности в результате модификации LTR. Термин «самоинактивирующие» (SIN) векторы относятся к дефектным по репликации векторам, например, в которых правая (3') область энхансер-промотора LTR, известная как область U3, была модифицирована (например, путем делеции или замены) для предотвращения вирусной транскрипции после первого раунда вирусной репликации. Дополнительное повышение безопасности обеспечивается заменой U3-области 5'-LTR гетерологичным промотором для управления транскрипцией вирусного генома во время производства вирусных частиц. Примеры гетерологичных промоторов, которые могут быть использованы, включают, например, промоторы обезьяньего вируса 40 (SV40) (например, ранний или поздний), цитомегаловируса (CMV) (например, немедленно ранний), вируса мышиного лейкоза Молони (MoMLV), вируса саркомы Рауса (RSV) и вируса простого герпеса (HSV) (тимидинкиназа).
Используемые в настоящей заявке термины «псевдотип» или «псевдотипирование» относятся к вирусу, белки вирусной оболочки которого были заменены белками другого вируса, обладающими предпочтительными характеристиками. Например, ВИЧ может быть псевдотипирован белками оболочки G-белка вируса везикулярного стоматита (VSV-G), который позволяет ВИЧ инфицировать более широкий круг клеток, поскольку белки оболочки ВИЧ (кодируемые геном env) обычно нацеливают вирус на CD4+презентирующие клетки.
В некоторых случаях лентивирусные векторы получают в соответствии с известными способами. См например, Kutner et al., BMC Biotechnol. 2009;9:10. doi: 10.1186/1472-6750-9-10; Kutner et al. Nat. Protoc. 2009;4(4):495-505. doi: 10.1038/nprot.2009.22.
Согласно определенным конкретным вариантам реализации, рассматриваемым в настоящей заявке, большинство или все последовательности основной цепи вирусного вектора получены из лентивируса, например, ВИЧ-1. Однако следует понимать, что можно использовать или комбинировать множество различных источников ретровирусных и/или лентивирусных последовательностей, и могут быть приспособлены многочисленные замены и изменения в некоторых из лентивирусных последовательностей без ухудшения способности вектора переноса выполнять описанные в настоящей заявке функции. Кроме того, в данной области известно множество лентивирусных векторов, см. Naldini et al., (1996a, 1996b и 1998); Zufferey et al., (1997); Dull et al., 1998, Патенты США №6013516; и 5994136, многие из которых могут быть адаптированы для получения вирусного вектора или переносящей плазмиды, рассматриваемых в настоящей заявке.
В различных вариантах реализации один или более полинуклеотидов, кодирующих вариант нуклеазы и/или донорную матрицу репарации, вводят в гемопоэтическую клетку путем трансдукции клетки аденовирусом, содержащим один или более полинуклеотидов.
Векторы на основе аденовируса способны к очень высокой эффективности трансдукции во многих типах клеток и не требуют деления клеток. С такими векторами были получены высокий титр и высокий уровень экспрессии. Указанный вектор может быть получен в больших количествах в относительно простой системе. Большинство аденовирусных векторов сконструированы так, что трансген заменяет гены Ad E1a, E1b и/или E3; впоследствии вектор с дефектом репликации размножается в клетках 293 человека, которые обеспечивают функции удаленного гена в транс-положении. Аденовирусные векторы могут трансдуцировать различные типы тканей in vivo, включая неделящиеся, дифференцированные клетки, такие как клетки печени, почек и мышц. Обычные аденовирусные векторы могут нести большую полезную нагрузку.
При генерировании и размножении современных аденовирусных векторов, которые имеют дефект по репликации, можно использовать уникальную линию клеток-хелперов, обозначенную 293, которая была трансформирована из эмбриональных клеток почки человека фрагментами ДНК Ad5 и конститутивно экспрессирует белки E1 (Graham et al., 1977). Поскольку область E3 можно удалить из генома аденовируса (Jones & Shenk, 1978), существующие векторы аденовируса, с помощью клеток 293, несут чужеродную ДНК в области E1, D3 или в обеих областях (Graham & Prevec, 1991). Аденовирусные векторы уже использовались для экспрессии генов эукариот (Levrero et al., 1991; Gomez-Foix et al., 1992) и разработки вакцин (Grunhaus & Horwitz, 1992; Graham & Prevec, 1992). Исследования по введению рекомбинантного аденовируса в разные ткани включают инстилляцию в трахею (Rosenfeld et al., 1991; Rosenfeld et al., 1992), инъекции в мышцы (Ragot et al., 1993), периферические внутривенные инъекции (Herz & Gerard, 1993) и стереотаксические инокуляция в головной мозг (Le Gal La Salle et al., 1993). Пример использования Ad-вектора в клиническом испытании включал полинуклеотидную терапию для противоопухолевой иммунизации с внутримышечной инъекцией (Sterman et al., Hum. Gene Ther. 7:1083-9 (1998)).
В различных вариантах реализации один или более полинуклеотидов, кодирующих вариант нуклеазы и/или донорную матрицу репарации, вводят в гемопоэтическую клетку путем трансдукции клетки вирусом простого герпеса, например, HSV-1, HSV-2, содержащим один или более полинуклеотидов.
Зрелый вирион HSV состоит из оболочечного икосаэдрического капсида с вирусным геномом, состоящим из линейной двухцепочечной молекулы ДНК размером 152 т.п.н. В одном варианте реализации вирусный вектор на основе HSV является дефицитным по одному или нескольким основным или второстепенным генам HSV. В одном варианте реализации вирусный вектор на основе HSV является дефектным по репликации. Большинство дефектных по репликации векторов HSV содержат делецию одного или нескольких промежуточно-ранних, ранних или поздних генов HSV для предотвращения репликации. Например, вектор HSV может быть дефектным по немедленно раннему гену, выбранному из группы, состоящей из: ICP4, ICP22, ICP27, ICP47 и их комбинации. Преимущества вектора HSV заключаются в его способности входить в латентную стадию, которая может привести к длительной экспрессии ДНК, и в его большом вирусном ДНК-геном, который может вмещать экзогенные вставки ДНК размером до 25 т.п.н. Векторы на основе HSV описаны, например, в патентах США. №№5837532, 5846782 и 5844513, а также в международных заявках на патент WO 91/02788, WO 96/04394, WO 98/15637 и WO 99/06583, каждая из которых полностью включена в настоящее описание посредством ссылки.
F. Клетки с отредактированным геномом
Клетки с отредактированным геномом, полученные способами, предусмотренными в частных вариантах реализации, содержат одну или более генных модификаций в гене PD-1 и позволяют создать улучшенные клеточные терапевтические средства для профилактики, лечения или ослабления по меньшей мере одного симптома рака, БТПХ, инфекционного заболевания, аутоиммунного заболевания, иммунодефицита или состояния ассоциированных с ними. Без привязки к какой-либо конкретной теории, мы полагаем, что композиции и способы, рассматриваемые в настоящей заявке, повышают эффективность адоптивной клеточной терапии, в частности, делая терапевтические клетки более устойчивыми к иммуносупрессивным сигналам и истощению.
Клетки с отредактированным геномом, рассматриваемые в частных вариантах реализации могут быть аутологичными/аутогенными («собственными») или неаутологичными («не-собственными», например, аллогенными, сингенными или ксеногенными). Термин «аутологичный» в настоящей заявке относится к клеткам от одного и того же субъекта. Термин «аллогенный», в настоящей заявке, относится к клеткам одного и того же биологического вида, которые генетически отличаются от сравниваемой клетки. Термин «сингенный» в настоящей заявке относится к клеткам другого субъекта, которые генетически идентичны сравниваемой клетке. Термин «ксеногенный», в настоящей заявке, относится к клеткам другого вида по сравнению со сравниваемой клеткой. В предпочтительных вариантах реализации клетки получают от субъекта-млекопитающего. В более предпочтительном варианте реализации клетки получают от субъекта-примата, необязательно - примата, не являющегося человеком. В наиболее предпочтительном варианте реализации клетки получают от человека.
Термин «изолированная клетка» относится к не встречающейся в природе клетке, например клетке, которая не существует в природе, модифицированной клетке, сконструированной клетке, рекомбинантной клетке и т.д., которая была получена из ткани или органа in vivo и по существу не содержит внеклеточного матрикса.
Используемый в настоящей заявке термин «популяция клеток» относится к множеству клеток, которые могут состоять из любого числа и/или комбинации гомогенных или гетерогенных типов клеток, как описано в другом месте в настоящей заявке. Например, для трансдукции Т-клеток популяция клеток может быть выделена или получена из периферической крови. Популяция клеток может содержать приблизительно 10%, приблизительно 20%, приблизительно 30%, приблизительно 40%, приблизительно 50%, приблизительно 60%, приблизительно 70%, приблизительно 80%, приблизительно 90% или приблизительно 100% типа клеток-мишеней для редактирования. В некоторых вариантах реализации Т-клетки могут быть выделены или очищены из популяции гетерогенных клеток с использованием способов, известных в данной области.
Иллюстративные примеры типов клеток, геном которых можно редактировать с использованием композиций и способов, предусмотренных в настоящей заявке, включают, среди прочего, клеточные линии, первичные клетки, стволовые клетки, клетки-предшественники и дифференцированные клетки и их смеси.
В предпочтительном варианте реализации композиции и способы редактирования генома используются для редактирования гемопоэтических клеток, более предпочтительно иммунных клеток и еще более предпочтительно Т-клеток.
Термины «Т-клетка» или «Т-лимфоцит» признаны в данной области и включают тимоциты, иммунные эффекторные клетки, регуляторные Т-клетки, наивные Т-лимфоциты, незрелые Т-лимфоциты, зрелые Т-лимфоциты, покоящиеся Т-лимфоциты или активированные Т-лимфоциты. Т-клетка может быть T-хелпером (Th), например T-хелпером 1 (Th1) или T-хелпером 2 (Th2). Т-клетка может быть хелперной T-клеткой (HTL; CD4+T-клеткой) CD4+T-клеткой, цитотоксической T-клеткой (CTL; CD8+T-клеткой), инфильтрирующей опухоль цитотоксической T-клеткой (TIL; CD8+T-клеткой), CD4+CD8+T клеткой, CD4- CD8- Т- клеткой или любой другой подгруппой Т-клеток. В одном варианте реализации Т-клетка представляет собой иммунную эффекторную клетку. В одном варианте реализации Т-клетка представляет собой NKT-клетку. Другие иллюстративные популяции Т-клеток, подходящие для использования в частных вариантах реализации, включают наивные Т-клетки и Т-клетки памяти.
В различных вариантах реализации клетки с отредактированным геномом содержат иммунные эффекторные клетки, содержащие ген PD-1, отредактированный композициями и способами, предусмотренными в настоящей заявке. «Иммунная эффекторная клетка» представляет собой любую клетку иммунной системы, которая выполняет одну или более эффекторных функций (например, цитотоксическая активность по уничтожению клеток, секреция цитокинов, индукция ADCC и/или CDC). Иллюстративные иммунные эффекторные клетки, рассматриваемые в частных вариантах реализации представляют собой T-лимфоциты, в частности цитотоксические T-клетки (CTL; CD8+T-клетки), TIL и хелперные T-клетки (HTL; CD4+T-клетки). В одном варианте реализации иммунные эффекторные клетки включают клетки природные киллеры (NK). В одном варианте реализации иммунные эффекторные клетки включают Т клетки-природные киллеры (NKT).
«Активные Т-клетки» и «молодые Т-клетки» используются взаимозаменяемо в частных вариантах реализации и относятся к фенотипам Т-клеток, когда Т-клетка способна к пролиферации и сопутствующему снижению дифференцировки. В частных вариантах реализации молодая Т-клетка имеет фенотип «наивной Т-клетки». В частных вариантах реализации молодые Т-клетки содержат один или более из или все из следующих биологических маркеров: CD62L, CCR7, CD28, CD27, CD122, CD127, CD197 и CD38. В одном варианте реализации молодые Т-клетки содержат один или более или все из следующих биологических маркеров: CD62L, CD127, CD197 и CD38. В одном варианте реализации молодые Т-клетки лишены экспрессии CD57, CD244, CD160, PD-1, CTLA4, PD-1 и LAG3.
Т-клетки могут быть получены из ряда источников, включая, среди прочего, мононуклеарные клетки периферической крови, костный мозг, ткань лимфатических узлов, пуповинную кровь, тимус, ткани из места инфекции, асцит, плевральный выпот, ткань селезенки и опухоли.
В частных вариантах реализации популяция клеток, включающая иммунные эффекторные клетки или Т-клетки, содержит отредактированный ген PD-1, где редактирование представляет собой DSB, репарированный с помощью NHEJ. В частных вариантах реализации иммунная эффекторная клетка или Т-клетка содержит отредактированный ген PD-1, где редактирование представляет собой DSB, репарированный NHEJ. В частных вариантах реализации редактирование представляет собой инсерцию или делецию (INDEL) приблизительно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14 15, 16, 17, 18., 19, 20, 21, 22, 23, 24, 25 или более нуклеотидов в кодирующей последовательности гена PD-1, предпочтительно в экзоне 5, экзоне 1 или экзоне 2 гена PD-1, более предпочтительно в SEQ ID NO: 25 (или SEQ ID NO: 27) в экзоне 5 гена PD-1, в SEQ ID NO: 30 (или SEQ ID NO: 32) в экзоне 1 гена PD-1 или в SEQ ID NO: 35 (или SEQ ID NO: 37) в экзоне 2 гена PD-1.
В частном варианте реализации редактирование представляет собой делецию+1, -1, -2, -3 или -4 нуклеотидов в кодирующей последовательности гена PD-1, предпочтительно в экзоне 5, экзоне 1 или экзоне 2 ген PD-1, более предпочтительно в SEQ ID NO: 25 (или SEQ ID NO: 27) в экзоне 5 гена PD-1, в SEQ ID NO: 30 (или SEQ ID NO: 32) в экзоне 1 ген PD-1 или SEQ ID NO: 35 (или SEQ ID NO: 37) в экзоне 2 гена PD-1.
В частных вариантах реализации популяция клеток, включающая иммунные эффекторные клетки или Т-клетки, содержит отредактированный ген PD-1, содержащий донорную матрицу репарации, встроенную в DSB, репарированный HDR.
В частных вариантах реализации популяция клеток, включающая иммунные эффекторные клетки или Т-клетки, содержит отредактированный ген PD-1, содержащий донорную матрицу репарации, содержащую ген PD-1 или его часть, и предназначена для введения одной или нескольких мутаций в геномную PD-1 последовательность с целью изменения экспрессии или передачи сигналов PD-1 и, предпочтительно, для уменьшения или устранения экспрессии и/или передачи сигналов PD-1.
В различных вариантах реализации клетка с отредактированным геномом содержит редактирование в гене PD-1 и дополнительно содержит полинуклеотид, кодирующий флип-рецептор PD-1, молекулу биспецифического Т-клеточного активатора (BiTE); цитокин (например, IL-2, инсулин, IFN-γ, IL-7, IL-21, IL-10, IL-12, IL-15 и TNF-α), хемокин (например, MIP-1α, MIP-1β, MCP-1, MCP-3 и RANTES), цитотоксин (например, перфорин, гранзим A и гранзим B), рецептор цитокинов (например, рецептор IL-2, рецептор IL-7, Рецептор IL-12, рецептор IL-15 и рецептор IL-21) или сконструированный антигенный рецептор (например, сконструированный T-клеточный рецептор (TCR), химерный антигенный рецептор (CAR), рецептор Daric или его компоненты, или химерный цитокиновый рецептор). В одном варианте реализации в клетку встраивают донорную матрицу репарации, содержащую полинуклеотид и вариант нуклеазы, и полинуклеотид встраивают в геном клетки в сайте DSB в гене PD-1 посредством HDR-репарации. Полинуклеотид также может быть введен в клетку в сайте, отличном от гена PD-1, например, путем трансдукции клетки вектором, содержащим полинуклеотид.
G. Композиции и составы
Композиции, рассматриваемые в частных вариантах реализации могут содержать один или более полипептидов, полинуклеотидов, векторов, содержащих их, а также композиции для редактирования генома и композиции с клеткой с отредактированным геномом, как предусмотрено в настоящей заявке. Композиции и способы редактирования генома, рассматриваемые в частных вариантах реализации полезны для редактирования сайта-мишени в гене человеческой запрограммированной клеточной гибели 1 (PD-1) в клетке или популяции клеток. В предпочтительных вариантах реализации композицию для редактирования генома используют для редактирования гена PD-1 в гемопоэтической клетке, например, Т-клетке или иммунной эффекторной клетке.
В различных вариантах реализации рассматриваемые в настоящей заявке композиции содержат вариант нуклеазы и, необязательно, фермент, изменяющий структуру концов, например, 3'-5' экзонуклеазу (Trex2). Вариант нуклеазы может быть в форме мРНК, которая вводится в клетку с помощью способов доставки полинуклеотидов, описанных выше, например, электропорации, липидных наночастиц и т.д. В одном варианте реализации композицию, содержащую мРНК, кодирующую вариант хоминг-эндонуклеазы или megaTAL. и, необязательно, 3'-5' экзонуклеазу, вводят в клетку с помощью способов доставки полинуклеотидов, описанных выше. Такая композиция может быть использована для создания клетки с отредактированным геномом или популяции клеток с отредактированным геномом клетку с помощью допускающего ошибки NHEJ.
В различных вариантах реализации рассматриваемые в настоящей заявке композиции содержат донорную матрицу репарации. Композиция может быть доставлена в клетку, которая экспрессирует или будет экспрессировать вариант нуклеазы, и, необязательно, фермент, изменяющий структуру концов. В одном варианте реализации композиция может быть доставлена в клетку, которая экспрессирует или будет экспрессировать вариант хоминг-эндонуклеазы или megaTAL и, необязательно, экзонуклеазу 3'-5'. Для создания клетки с отредактированным геномом или популяции клеток с отредактированным геномом может быть использована экспрессия редактирующих ген ферментов в присутствии донорной матрицы репарации методом HDR.
В частных вариантах реализации рассматриваемые в настоящей заявке композиции содержат популяцию клеток, вариант нуклеазы и, необязательно, донорную матрицу репарации. В частных вариантах реализации композиции, рассматриваемые в настоящей заявке, содержат популяцию клеток, вариант нуклеазы, фермент, изменяющий структуру концов и, необязательно, донорную матрицу репарации. Вариант нуклеазы и/или фермент, изменяющий структуру концов, может быть в форме мРНК, которая введена в клетку с помощью способов доставки полинуклеотидов, описанных выше.
В частных вариантах реализации рассматриваемые в настоящей заявке композиции содержат популяцию клеток, вариант хоминг-эндонуклеазы или megaTAL и, необязательно, донорную матрицу репарации. В частных вариантах реализации рассматриваемые в настоящей заявке композиции содержат популяцию клеток, вариант хоминг-эндонуклеазы или megaTAL, экзонуклеазу 3'-5' и, необязательно, донорную матрицу репарации. Вариант хоминг-эндонуклеазы, megaTAL и/или 3'-5' экзонуклеаза может быть в форме мРНК, которая введена в клетку с помощью способов доставки полинуклеотидов, описанных выше.
В частных вариантах реализации популяция клеток содержит генетически модифицированные иммунные эффекторные клетки.
Композиции включают, среди прочего, фармацевтические композиции. «Фармацевтическая композиция» относится к композиции, составленной в виде фармацевтически приемлемых или физиологически приемлемых растворов для введения в клетку или животному, либо отдельно, либо в комбинации с одним или несколькими другими видами терапии. Также следует понимать, что при желании композиции можно вводить в комбинации с другими агентами, такими как, например, цитокины, факторы роста, гормоны, малые молекулы, химиотерапевтические средства, пролекарства, лекарства, антитела или другие. различные фармацевтически активные вещества. Для других компонентов, которые также могут быть включены в композиции, практически нет ограничений при условии, что дополнительные агенты не оказывают вредного влияния на композицию.
Фраза «фармацевтически приемлемый» используется в настоящей заявке для обозначения тех соединений, материалов, композиций и/или дозированных форм, которые в рамках здравого медицинского суждения подходят для использования в контакте с тканями людей и животных без чрезмерной токсичности, раздражения, аллергического ответа или других проблем или осложнений, соизмеримых с разумным соотношением пользы/риска.
Термин «фармацевтически приемлемый носитель» относится к разбавителю, адъюванту, вспомогательному веществу или носителю, с которым вводят терапевтические клетки. Иллюстративными примерами фармацевтических носителей могут быть стерильные жидкости, такие как среды для культивирования клеток, вода и масла, включая масла нефтяного, животного, растительного или синтетического происхождения, такие как арахисовое масло, соевое масло, минеральное масло, кунжутное масло и тому подобное. Солевые растворы и водные растворы декстрозы и глицерина также могут быть использованы в качестве жидких носителей, особенно для инъекционных растворов. Подходящие фармацевтические вспомогательные вещества в частных вариантах реализации включают крахмал, глюкозу, лактозу, сахарозу, желатин, солод, рис, муку, мел, силикагель, стеарат натрия, моностеарат глицерина, тальк, хлорид натрия, сухое обезжиренное молоко, глицерин, пропилен, гликоль, воду, этанол и тому подобное. За исключением случаев, когда какой-либо обычный носитель или агент несовместим с активным ингредиентом, предполагается его использование в таких терапевтических композициях. В композиции также могут быть включены дополнительные активные ингредиенты.
В одном варианте реализации композиция, содержащая фармацевтически приемлемый носитель, является пригодной для введения субъекту. В частных вариантах реализации композиция, содержащая носитель, пригодна для парентерального введения, например, внутрисосудистого (внутривенного или внутриартериального), внутрибрюшинного или внутримышечного введения. В частных вариантах реализации композиция, содержащая фармацевтически приемлемый носитель, пригодна для внутрижелудочкового, интраспинального или интратекального введения. Фармацевтически приемлемые носители включают стерильные водные растворы, среды для культивирования клеток или дисперсии. Использование таких сред и агентов для фармацевтически активных веществ хорошо известно в данной области. За исключением случаев, когда какой-либо обычный носитель или агент несовместим с трансдуцированными клетками, предполагается его применение в таких фармацевтических композициях.
В частных вариантах реализации композиции, рассматриваемые в настоящей заявке, содержат генетически модифицированные Т-клетки и фармацевтически приемлемый носитель. Композицию, содержащую композицию на основе клеток, рассматриваемая в настоящей заявке, можно вводить отдельно посредством энтерального или парентерального способов введения или в комбинации с другими подходящими соединениями для достижения желаемых целей лечения.
Фармацевтически приемлемый носитель должен иметь достаточно высокую чистоту и достаточно низкую токсичность, чтобы быть пригодным для введения человеку, подвергаемому лечению. Кроме того, он должен поддерживать или увеличивать стабильность композиции. Фармацевтически приемлемый носитель может быть жидким или твердым, и его выбирают с учетом планируемого способа введения для обеспечения желаемой массы, консистенции и т.д. при сочетании с другими компонентами композиции. Например, фармацевтически приемлемым носителем может быть без ограничения связующий агент (например, предварительно желатинизированный кукурузный крахмал, поливинилпирролидон или гидроксипропилметилцеллюлоза и др.), наполнитель (например, лактоза и другие сахара, микрокристаллическая целлюлоза, пектин, желатин, сульфат кальция, этилцеллюлоза, полиакрилаты, гидрофосфат кальция и др.), скользящее вещество (например, стеарат магния, тальк, диоксид кремния, коллоидный диоксид кремния, стеариновая кислота, стеараты металлов, гидрогенизированные растительные масла, кукурузный крахмал, полиэтиленгликоли, бензоат натрия, ацетат натрия и т.д.), дезинтегрант (например, крахмал крахмалгликолят натрия и т.д.) или смачивающий агент (например, лаурилсульфат натрия и т.д.). Другие подходящие фармацевтически приемлемые носители для рассматриваемых в настоящей заявке композиций включают, среди прочего, воду, солевые растворы, спирты, полиэтиленгликоли, желатины, амилозы, стеараты магния, тальки, кремниевые кислоты, вязкие парафины, гидроксиметилцеллюлозы, поливинилпирролидоны и тому подобное.
Такие растворы-носители также могут содержать буферы, разбавители и другие подходящие добавки. Используемый в настоящей заявке термин «буфер» относится к раствору или жидкости, химический состав которых нейтрализует кислоты или основания без значительного изменения pH. Примеры буферов, рассматриваемых в настоящей заявке, включают, среди прочего, забуференный фосфатом физиологический раствор (PBS) Дульбекко, раствор Рингера, 5% декстрозу в воде (D5W), нормальный/физиологический солевой раствор (0,9% NaCl).
Фармацевтически приемлемые носители могут присутствовать в количествах, достаточных для поддержания рН композиции приблизительно 7. В альтернативном варианте композиция имеет рН в диапазоне от приблизительно 6,8 до приблизительно 7,4, например, 6,8, 6,9, 7,0, 7,1, 7,2. 7.3 и 7.4. В еще одном варианте реализации композиция имеет рН приблизительно 7,4.
Композиции, рассматриваемые в настоящей заявке, могут содержать нетоксичную фармацевтически приемлемую среду. Композиции могут быть суспензией. Используемый в настоящей заявке термин «суспензия» относится к условиям без слипания, в которых клетки не прикреплены к твердой подложке. Например, клетки, поддерживаемые в виде суспензии, можно взбалтывать или перемешивать без прилипания к подложке, такой как культуральная чашка.
В частных вариантах реализации композиции, рассматриваемые в настоящей заявке, составлены в виде суспензии, в которой Т-клетки с отредактированным геномом диспергированы в приемлемой жидкой среде или растворе, например, в солевой или бессывороточной среде, в пакете для внутривенного (IV) вливания или тому подобном. Приемлемые разбавители включают, среди прочего, воду, PlasmaLyte, раствор Рингера, изотонический раствор хлорида натрия (физиологический раствор), бессывороточную среду для культивирования клеток и среду, пригодную для криогенного хранения, например, среду Cryostor®.
В некоторых вариантах реализации фармацевтически приемлемый носитель по существу не содержит природных белков человеческого или животного происхождения и пригоден для хранения композиции, содержащей популяцию Т-клеток с отредактированным геномом. Терапевтическая композиция предназначена для введения пациенту-человеку и, поэтому, по существу не содержит компонентов клеточной культуры, таких как бычий сывороточный альбумин, конская сыворотка и фетальная бычья сыворотка.
В некоторых вариантах реализации композиции составлены в фармацевтически приемлемой среде для культивирования клеток. Такие композиции пригодны для введения субъектам-людям. В частных вариантах реализации фармацевтически приемлемая среда для культивирования клеток представляет собой бессывороточную среду.
Среда, не содержащая сыворотки, имеет несколько преимуществ перед средой, содержащей сыворотку, включая упрощенный и более четко определенный состав, меньшую степень загрязнения, устранение потенциального источника инфекционных агентов и более низкую стоимость. В различных вариантах реализации бессывороточная среда является не содержащей компонентов животного происхождения и необязательно безбелковой. Необязательно, среда может содержать биофармацевтически приемлемые рекомбинантные белки. Термин «не содержащая компонентов животного происхождения» среда относится к среде, в которой компоненты получены из не животных источников. В среде без компонентов животного происхождения рекомбинантные белки заменяют нативные животные белки, а питательные вещества получают из синтетических, растительных или микробных источников. «Безбелковая» среда, напротив, определяется как по существу не содержащая белка.
Иллюстративные примеры бессывороточных сред, используемых в конкретных композициях, включают, среди прочего, QBSF-60 (Quality Biological, Inc.), StemPro-34 (Life Technologies) и X-VIVO 10.
В предпочтительном варианте реализации композиции, содержащие Т-клетки с отредактированным геномом, составлены в растворе PlasmaLyte.
В различных вариантах реализации композиции, содержащие Т-клетки с отредактированным геномом, готовят в криоконсервирующей среде. Например, криоконсервирующую среду с криоконсервационными агентами можно использовать для поддержания высокого выхода жизнеспособных клеток после оттаивания. Иллюстративные примеры криоконсервирующих сред, используемых в конкретных композициях, включают, среди прочего, CryoStor CS10, CryoStor CS5 и CryoStor CS2.
В одном варианте реализации композиции составляют в растворе, содержащем смесь 50:50 PlasmaLyte A с CryoStor CS10.
В частных вариантах реализации композиция по существу не содержит микоплазмы, эндотоксина и микробных загрязнений. Под «по существу не содержат» в отношении эндотоксина подразумевается, что на дозу клеток приходится меньше эндотоксина, чем разрешено FDA для биопрепарата, то есть суммарное количество эндотоксина 5 EU/кг массы тела в день, что в среднем на человека весом 70 кг составляет 350 EU на суммарную дозу клеток. В частных вариантах реализации композиции, содержащие гемопоэтические стволовые или прогениторные клетки, трансдуцированные ретровирусным вектором, рассматриваемым в настоящей заявке, содержат от приблизительно 0,5 EU/мл до приблизительно 5,0 EU/мл или приблизительно 0,5 EU/мл, 1,0 EU/мл, 1,5 EU/мл, 2,0 EU/мл, 2,5 EU/мл, 3,0 EU/мл, 3,5 EU/мл, 4,0 EU/мл, 4,5 EU/мл или 5,0 EU/мл.
В некоторых вариантах реализации предложены композиции и составы, подходящие для доставки полинуклеотидов, включая, среди прочего, одну или более мРНК, кодирующих одну или более перепрограммированных нуклеаз, и, необязательно, ферменты, изменяющие структуру концов.
Иллюстративные составы для доставки ex vivo могут также включать применение различных трансфекционных агентов, известных в данной области, таких как фосфат кальция, электропорация, тепловой шок и различные липосомные составы (то есть для трансфекции, опосредованной липидами). Липосомы, как описано более подробно ниже, представляют собой липидные бислои, захватывающие фракцию водной жидкости. ДНК самопроизвольно связывается с внешней поверхностью катионных липосом (в силу своего заряда), и эти липосомы будут взаимодействовать с клеточной мембраной.
В частных вариантах реализации составление фармацевтически приемлемых растворов носителей хорошо известно специалистам в данной области, как и разработка подходящих режимов дозирования и лечения для применения конкретных композиций, описанных в настоящей заявке, в различных схемах лечения, включая, например, энтеральное и парентеральное, например, внутрисосудистое, внутривенное, внутрисердечное, внутрикостное, внутрижелудочковое, внутримозговое, внутричерепное, интраспинальное, интратекальное и интрамедуллярное введение и состав. Специалисту в данной области будет понятно, что конкретные варианты реализации, рассматриваемые в настоящей заявке, могут содержать другие составы, такие как те, которые хорошо известны в области фармацевтики, и описаны, например, в справочнике Remington: The Science and Practice of Pharmacy, т.I и т.II. 22-е издание, под редакцией Loyd V. Allen Jr. Philadelphia, PA: Pharmaceutical Press; 2012, который включен в настоящую заявку посредством ссылки во всей своей полноте.
H. Методы лечения клетками с отредактированным геномом
Клетки с отредактированным геномом, изготовленные с помощью композиций и способов, предусмотренных в настоящей заявке, дают улучшенные лекарственные продукты для применения при профилактике, лечении или ослаблении по меньшей мере одного симптома рака, БТПХ, инфекционного заболевания, аутоиммунного заболевания, воспалительного заболевания или иммунодефицита. Используемый в настоящей заявке термин «лекарственный продукт» относится к генетически модифицированным клеткам, полученным с применением композиций и способов, предусмотренных в настоящей заявке. В частных вариантах реализации лекарственный продукт содержит генетически отредактированные иммунные эффекторные клетки или Т-клетки. Кроме того, Т-клетки с отредактированным геномом, рассматриваемые в частных вариантах реализации обеспечивают более безопасную и более эффективную адоптивную клеточную терапию, поскольку они устойчивы к истощению Т-клеток и демонстрируют повышенную стойкость и жизнестойкость в микроокружении опухоли, что может позволить продолжительную терапию.
В частных вариантах реализации эффективное количество иммунных эффекторных клеток или Т-клеток с отредактированным геномом, содержащих отредактированный ген PD-1, вводят субъекту для предотвращения, лечения или ослабления по меньшей мере одного симптома рака, БТПХ, инфекционного заболевания, аутоиммунного заболевания, воспалительного заболевания или иммунодефицита.
В частных вариантах реализации отредактированные PD-1 клетки по существу или полностью не экспрессируют PD-1 и, следовательно, не имеют или по существу не имеют функциональной экспрессии PD-1, например, не обладают способностью усиливать истощение Т-клеток и ингибировать экспрессию провоспалительных цитокинов. В частных вариантах реализации иммунные эффекторные клетки с отредактированным геномом, в которых отсутствует PD-1, являются более устойчивыми к иммуносупрессивным сигналам от микроокружения опухоли и проявляют повышенную жизнестойкость и устойчивость к истощению Т-клеток.
В частных вариантах реализации способ предотвращения, лечения или ослабления по меньшей мере одного симптома рака включает введение субъекту эффективного количества иммунных эффекторных клеток или Т-клеток с отредактированным геномом, содержащих отредактированный ген PD-1 и сконструированный TCR, CAR, или Daric, или другой терапевтический трансген для перенацеливания этих клеток на опухоль или рак. Генетически модифицированные клетки представляют собой более длительный и устойчивый лекарственный продукт, поскольку эти клетки более устойчивы к иммуносупрессивным сигналам из микроокружения опухоли благодаря редактированию гена PD-1 для уменьшения или устранения экспрессии PD-1.
В частных вариантах реализации клетки с отредактированным геномом, рассматриваемые в настоящей заявке, используют при лечении солидных опухолей или раковых заболеваний.
В частных вариантах реализации клетки с отредактированным геномом, рассматриваемые в настоящей заявке, используют при лечении солидных опухолей или раковых заболеваний, включая, среди прочего, следующие: рак надпочечника, адренокортикальная карцинома, рак анального канала, рак червеобразного отростка, астроцитома, атипичная тератоидная/рабдоидная опухоль, базальноклеточный рак, рак желчных протоков, рак мочевого пузыря, рак кости, рак головного мозга/ЦНС, рак молочной железы, опухоли бронхов, опухоли сердца, рак шейки матки, холангиокарцинома, хондросаркома, хордом, рак толстой кишки, колоректальный рак, краниофарингиома, протоковая карцинома (DCIS), рак эндометрия, эпендимома, рак пищевода, эстезионейробластома, саркома Юинга, экстракраниальна герминома, внегонадная герминома, рак глаза, рак фаллопиевых труб, фиброзная гистиосаркома, фибросаркома, рак желчного пузыря, рак желудка, желудочно-кишечные карциноидные опухоли, желудочно-кишечная стромальная опухоль (GIST), опухоли зародышевых клеток, глиома, глиобластома, рак головы и шеи, гемангиобластома, гепатоцеллюлярный рак, рак гипофарингеального отдела, интраокулярная меланома, саркома Капоши, рак почки, рак гортани, лейомиосаркома, рак губы, липосаркома, рак печени, рак легкого, немелкоклеточный рак легкого, рак легкого, злокачественная мезотелиома, медуллярная карцинома, медуллобластома, менангиома, меланома, карцинома Меркеля, срединная карцинома, рак ротовой полости, миксосаркома, миелодиспластический синдром, миелопролиферативные новообразования, рак полости носа и околоносовых пазух, рак носоглотки, нейробластома, олигодендроглиома, рак ротовой полости, рак полости рта, рак ротоглотки, остеосаркома, рак яичников, рак поджелудочной железы, опухоли островковых клеток поджелудочной железы, папиллярный рак, параганглиома, рак паращитовидной железы, рак полового члена, рак глотки, феохромоцитома, пинеалома, опухоль гипофиза, плевропульмональная бластома, первичный рак брюшины, рак простаты, рак прямой кишки, ретинобластома, почечно-клеточный рак, почечной лоханки и таза, рабдомиосаркома, рак слюнных желез, карцинома сальных желез, рак кожи, саркома мягких тканей, плоскоклеточный рак, мелкоклеточный рак легкого, рак тонкой кишки, рак желудка, карцинома потовых желез, синовиома, рак яичка, рак горла, рак тимуса, рак щитовидной железы, рак уретры, рак матки, саркома матки, рак влагалища, рак сосудов, рак вульвы и опухоль Вильмса.
В частных вариантах реализации клетки с отредактированным геномом, рассматриваемые в настоящей заявке, используются при лечении солидных опухолей или раковых заболеваний, включая, без ограничения, рак печени, рак поджелудочной железы, рак легких, рак молочной железы, рак мочевого пузыря, рак мозга, рак кости, рак щитовидной железы, рак почки или рак кожи.
В частных вариантах реализации рассматриваемые в настоящей заявке клетки с отредактированным геномом используют для лечения различных видов рака, включая, среди прочего, рак поджелудочной железы, мочевого пузыря и легкого.
В частных вариантах реализации клетки с отредактированным геномом, рассматриваемые в настоящей заявке, используют для лечения гемобластозов или гематологических раковых заболеваний.
В частных вариантах реализации клетки с отредактированным геномом, рассматриваемые в настоящей заявке, используют для лечения В-клеточных злокачественных новообразований, включая, среди прочего, лейкозы, лимфомы и множественную миелому.
В частных вариантах реализации клетки с отредактированным геномом, рассматриваемые в настоящей заявке, используют для лечения гемобластозов, включая, среди прочего, лейкозы, лимфомы и множественные миеломы: острый лимфоцитарный лейкоз (ОЛЛ), острый миелоидный лейкоз (ОМЛ), миелобластный, промиелоцитарный, миеломоноцитарный, моноцитарный, эритролейкоз, лейкоз ворсистых клеток (ЛВК), хронический лимфоцитарный лейкоз (ХЛЛ) и хронический миелоидный лейкоз (ХМЛ), хронический миеломоноцитарный лейкоз (ХММЛ) и истинную полицитемию, лимфому Ходжкина, лимфому Ходжкина с преобладанием узловых лимфоцитов, лимфому Беркитта, лимфоцитарную лимфому (SLL), диффузную В-крупноклеточную лимфому, фолликулярную лимфому, крупноклеточную иммунобластную лимфому, B-лимфобластную лимфому из клеток-предшественников, лимфому из мантийных клеток, лимфому маргинальной зоны, фунгоидный микоз, анапластическую крупноклеточную лимфому, синдром Сезари, Т-лимфобластную лимфому из клеток-предшественников, множественную миелому, явную множественную миелому, вялотекущую множественную миелому, плазмоклеточный лейкоз, несекреторную миелому, миелому IgD, остеосклеротическую миелому, одиночную плазмоцитому кости и экстрамедуллярную плазмоцитому.
Предпочтительные клетки для применения в способах редактирования генома, рассматриваемых в настоящей заявке, включают аутологичные/аутогенные («собственные») клетки, предпочтительно гемопоэтические клетки, более предпочтительно Т-клетки и более предпочтительно иммунные эффекторные клетки или клетки Treg.
В частных вариантах реализации предлагаются способы, включающие введение терапевтически эффективного количества клеток с отредактированным геномом, рассматриваемых в настоящей заявке, или композиции, содержащей их, нуждающемуся в этом пациенту, в качестве монотерапии или в сочетании с одним или несколькими терапевтическими агентами. По некоторым данным, такие клетки используют при лечении пациентов с риском развития рака, БТПХ, инфекционного заболевания, аутоиммунного заболевания, воспалительного заболевания или иммунодефицита. Таким образом, конкретные варианты реализации включают лечение, профилактику или ослабление по меньшей мере одного симптома рака, инфекционного заболевания, аутоиммунного заболевания, воспалительного заболевания или иммунодефицита, включающие введение субъекту, нуждающемуся в этом, терапевтически эффективного количества клеток с отредактированным геномом, рассматриваемых в настоящей заявке.
В одном варианте реализации способ лечения рака, БТПХ, инфекционного заболевания, аутоиммунного заболевания, воспалительного заболевания или иммунодефицита у нуждающегося в этом субъекта включает введение эффективного количества, например, терапевтически эффективного количества композиции, содержащей клетки с отредактированным геномом, рассматриваемые в настоящей заявке. Количество и частота введения будут определяться такими факторами, как состояние пациента, а также тип и тяжесть заболевания пациента, хотя надлежащие дозы могут быть определены клиническими испытаниями.
В одном иллюстративном варианте реализации эффективное количество клеток с отредактированным геномом, предоставленное субъекту, составляет по меньшей мере 2 х 106 клеток/кг, по меньшей мере 3 х 106 клеток/кг, по меньшей мере 4 х 106 клеток/кг, по меньшей мере 5 х 106 клеток/кг, по меньшей мере 6 х 106 клеток/кг, по меньшей мере 7 х 106 клеток/кг, по меньшей мере 8 х 106 клеток/кг, по меньшей мере 9 х 106 клеток/кг или, по меньшей мере 10 х 106 клеток/кг, или более клеток/кг, включая все промежуточные дозы клеток.
В другом иллюстративном варианте реализации эффективное количество клеток с отредактированным геномом, предоставленное субъекту, составляет приблизительно 2 x 106 клеток/кг, приблизительно 3 x 106 клеток/кг, приблизительно 4 x 106 клеток/кг, приблизительно 5 x 106 клеток/кг, приблизительно 6 x 106 клеток/кг, приблизительно 7 x 106 клеток/кг, приблизительно 8 x 106 клеток/кг, приблизительно 9 x 106 клеток/кг, или приблизительно 10 x 106 клеток/кг, или более клеток/кг, включая все промежуточные дозы клеток.
В другом иллюстративном варианте реализации эффективное количество клеток с отредактированным геномом, предоставленное субъекту, составляет от приблизительно 2 х 106 клеток/кг до приблизительно 10 х 106 клеток/кг, от приблизительно 3 х 106 клеток/кг до приблизительно 10 х 106 клеток/кг, от приблизительно 4 х 106 клеток/кг до приблизительно 10 х 106 клеток/кг, от приблизительно 5 х 106 клеток/кг до приблизительно 10 х 106 клеток/кг, от 2 х 106 клеток/кг до приблизительно 6 х 106 клеток/кг, от 2 х 106 клеток/кг до приблизительно 7 × 106 клеток/кг, от 2 × 106 клеток/кг до приблизительно 8 × 106 клеток/кг, от 3 × 106 клеток/кг до приблизительно 6 × 106 клеток/кг, от 3 × 106 клеток/кг до от приблизительно 7 х 106 клеток/кг, от 3 х 106 клеток/кг до приблизительно 8 х 106 клеток/кг, от 4 х 106 клеток/кг до приблизительно 6 х 106 клеток/кг, от 4 х 106 клеток/кг до приблизительно 7 х 106 клеток/кг, от 4 х 106 клеток/кг до приблизительно 8 х 106 клеток/кг, от 5 х 106 клеток/кг до приблизительно 6 х 106 клеток/кг, от 5 х 106 клеток/кг до приблизительно 7 х 106 клеток/кг, от 5 х 106 клеток/кг до приблизительно 8 х 106 клеток/кг или от 6 х 106 клеток/кг до приблизительно 8 х 106 клеток/кг, включая все промежуточные дозы клеток.
Специалист в данной области техники поймет, что для осуществления желаемой терапии может потребоваться многократное введение композиций, рассматриваемых в частных вариантах реализации. Например, композицию можно вводить 1, 2, 3, 4, 5, 6, 7, 8, 9 или 10 или более раз в течение 1 недели, 2 недель, 3 недель, 1 месяца, 2 месяцев, 3 месяца, 4 месяца, 5 месяцев, 6 месяцев, 1 год, 2 года, 5 лет, 10 лет или более.
В некоторых вариантах реализации может быть желательным вводить активированные Т-клетки субъекту, а затем впоследствии повторно забирать у него кровь (или проводить аферез), активировать Т-клетки из нее и повторно вводить пациенту эти активированные и размноженные Т-клетки. Указанный процесс можно выполнять несколько раз каждые несколько недель. В некоторых случаях можно использовать Т-клетки из взятой крови объемом от 10 до 400 см3. В некоторых вариантах реализации активируют Т-клетки из взятой крови объемом 20, 30, 40, 50, 60, 70, 80, 90, 100, 150, 200, 250, 300, 350 или 400 см 3 или более. Без привязки к какой-либо конкретной теории, применение такого протокола множественного взятия крови/множественной реинфузии может служить для отбора определенных популяций Т-клеток.
Введение композиций, рассматриваемых в частных вариантах реализации можно осуществлять любым удобным способом, включая ингаляцию аэрозоля, инъекцию, прием внутрь, трансфузию, имплантацию или трансплантацию. В предпочтительном варианте реализации композиции вводят парентерально. Фразы «парентеральное введение» и «введенный парентеральное», используемые в настоящей заявке, относятся к способам введения, отличным от энтерального и местного введения, обычно путем инъекции, и включают, без ограничения, внутрисосудистые, внутривенные, внутримышечные, внутриартериальные, интратекальные, внутрикапсулярные, интраорбитальные, внутриопухолевые, внутрисердечные, внутрикожные, внутрибрюшинные, транстрахеальные, подкожные, субкутикулярные, внутрисуставные, субкапсулярные, субарахноидальные, внутриспинальные и интрастернальные инъекции и инфузии. В одном варианте реализации композиции, рассматриваемые в настоящей заявке, вводят субъекту путем прямой инъекции в опухоль, лимфатический узел или участок инфекции.
В одном варианте реализации способ лечения субъекта, у которого диагностирован рак, включает удаление иммунных эффекторных клеток у субъекта, редактирование генома указанных иммунных эффекторных клеток и получение популяции геномом иммунных эффекторных клеток с отредактированным, и введение популяции иммунных эффекторных клеток с отредактированным геномом тому же субъекту. В предпочтительном варианте реализации иммунные эффекторные клетки включают Т-клетки.
Способы введения клеточных композиций, рассматриваемые в частных вариантах реализации включают любой способ, который эффективен для того, чтобы привести к реинтродукции ex vivo иммунных эффекторных клеток с отредактированным геномом или к реинтродукции предшественников иммунных эффекторных клеток с отредактированным геномом, которые при введении субъекту дифференцируются в зрелые иммунные эффекторные клетки. Один метод включает редактирование генома Т-клеток периферической крови ex vivo и возвращение этих трансдуцированных клеток субъекту.
Все публикации, патентные заявки и выданные патенты, процитированные в описании, включены в настоящую заявку посредством ссылки, как если бы каждая отдельная публикация, патентная заявка или выданный патент были прямо и индивидуально указаны как включенные в качестве ссылки.
Хотя для целей ясности понимания вышеизложенные варианты реализации были описаны в некоторых деталях в качестве иллюстрации и примера, для специалиста в данной области техники будет вполне очевидно в свете идей, рассматриваемых в настоящей заявке, что могут быть сделаны некоторые изменения и модификации без отступления от сущности или объема прилагаемой формулы изобретения. Следующие примеры приведены только для иллюстрации, а не для ограничения. Специалисты в данной области техники легко распознают множество некритических параметров, которые могут быть изменены или модифицированы для получения в основном аналогичных результатов.
ПРИМЕРЫ
Пример 1
Перепрограммирование I-OnuI для разрушения внутриклеточного сигнального мотива в гене рецептора запрограммированной смерти-1 (pd-1)
PD-1 экспрессируется на плазматической мембране Т-клеток после стимуляции и активации антигенных рецепторов. PD-1 содержит сигнальный пептид, внеклеточный IgV-подобный домен, трансмембранный домен и внутриклеточный хвост, который содержит мотив иммунорецептора на основе тирозина (ITIM, консенсусная последовательность S/I/V/LxYxxI/V/L) и иммунорецепторный мотив переключения на основе тирозина (ITSM, консенсусная последовательность TxYxxV/I). Рисунок 1А. Тирозин в аминокислотном положении 248 в ITSM PD-1 становится фосфорилированным при связывании лиганда PD-L1/2 одновременно с активацией Т-клеток, создавая субстрат связывания для белка, содержащего домен SH2, тирозин-фосфатазы-2 (SHP2, см. Chemnitz JM). et. al. J. Immunol. 2004 Jul 15; 173 (2): 945-54.). Рекрутинг SHP2 в плазматическую мембрану противодействует управляемым фосфотирозином сигналам активации в Т-клетках (Yokosuka T et al., J Exp Med. 2012 Jun 4; 209 (6): 1201-17) и подавляет длительность активированного состояния Т-клеток. Кодон для фосфорилированного ITSM тирозина охвачен каноническим «центральным- 4» мотивом расщепления I-OnuI, ATAC. Фигура 1B. Был разработан вариант хоминг-эндонуклеазы, нацеленный на последовательность-мишень 22 п.н. (SEQ ID NO: 25), центрированную на указанном центральном -4 мотиве в экзоне 5 гена PD-1.
Без привязки к какой-либо конкретной теории, предполагается, что все возможные события инсерции/делации («инделы») в «центральной-4» последовательности АТАС и вблизи от нее полностью уничтожат один или более существенных признаков мотива ITSM, причем наиболее возможные инделы уничтожат сам кодон тирозина-248. Эти инделы, вероятно, будут генерировать доминантно-отрицательные белки PD-1, содержащие нормальный внеклеточный домен и нефункциональный внутриклеточный сигнальный домен. Доминирующие отрицательные белки PD-1 могут действовать как «поглотитель» для лигандов PD-1, тем самым уменьшая или устраняя иммуносупрессивную передачу сигналов. Кроме того, поскольку активированные Т-клетки усиливают экспрессию PD-L1; поскольку взаимодействия PD-1:PD-L1 происходят среди Т-клеток, как в цис-, так и в транс-положении; и поскольку эти взаимодействия важны для функции Т-клеток, редактирование генов для нарушения передачи сигналов PD-1 без влияния на экспрессию может сохранить возможные управляемые PD-L1 функции.
Таким образом, I-OnuI был перепрограммирован для нацеливания на кодирующую область ITSM путем конструирования модульных библиотек, содержащих вариабельные аминокислотные остатки в интерфейсе распознавания ДНК. Для конструирования этих вариантов вырожденные кодоны встраивали в ДНК-связывающие домены I-OnuI с использованием олигонуклеотидов. Олигонуклеотиды, кодирующие эти вырожденные кодоны, использовали в качестве ПЦР-матриц для создания библиотек вариантов путем рекомбинации разрыва в штамме дрожжей S. cerevisiae. Каждая библиотека вариантов охватывала N- либо С-концевой домен распознавания ДНК I-OnuI и содержала ~ 107-108 уникальных трансформантов. Полученные библиотеки поверхностных дисплеев подвергали скринингу с помощью проточной цитометрии на активность расщепления в отношении сайта-мишени, содержащего «полусайты» соответствующих доменов (SEQ ID NO: 28-29), как показано на фиг.2.
Дрожжи, имеющие HE I-OnuI с перепрограммированными N- и C-концевым доменом, очищали и экстрагировали плазмидную ДНК. Проводили ПЦР-реакции для амплификации перепрограммированных доменов, которые впоследствии трансформировали в S. cerevisiae для создания библиотеки комбинаций перепрограммированных доменов. Из этой библиотеки идентифицировали полностью перепрограммированные варианты I-OnuI, которые распознают полный сайт-мишень (SEQ ID NO: 25), присутствующий в кодирующей области ITSM в экзоне 5 гена PD-1, и очищали их.
Активность перепрограммированных I-OnuI HE, которые нацелены на область кодирования ITSM PD-1 в экзоне 5, измеряли с использованием хромосомно интегрированной флуоресцентной репортерной системы (Certo et. Al., 2011). Полностью перепрограммированные I-OnuI HE, которые связывают и расщепляют последовательность-мишень PD-1 ITSM, клонировали в плазмиды экспрессии млекопитающих и затем индивидуально трансфицировали в клеточную линию фибробластов HEK 293T, содержащую последовательность-мишень экзона5 PD-1, расположенную в направлении против хода транскрипции от гена вне рамки трансляции, кодирующего флуоресцентный белок mCherry. Расщепление встроенного сайта-мишень указанной HE и накопление инделов после репарации ДНК посредством негомологичного соединения концов (NHEJ) приводит к тому, что приблизительно один из трех репарированных локусов помещает флуоресцентный репортерный ген обратно «в рамку». Поэтому процентное содержание клеток HEK 293T с флуоресцирующим mCherry используют в качестве показателя активности эндонуклеазы в хромосомно встроенной последовательности-мишени. Полностью перепрограммированный I-OnuI HE (PD-1.ITSM.ex5_RD1_CV3-08, SEQ ID NO: 6), нацеленный на сайт экзона 5 PD-1, показал очень умеренную эффективность экспрессии mCherry в клеточном хромосомном контексте. Фигура 3А. Вариант HE имел умеренные свойства аффинности к ДНК при измерении с помощью равновесного титрования субстрата (фиг.3 В).
Затем была сгенерирована библиотека вторичных вариантов I-OnuI путем выполнения случайного мутагенеза в варианте PD-1.ITSM.ex5_RD1_CV3-08 HE. Чтобы выделить варианты с улучшенной каталитической эффективностью, выполняли сортировку потока на основе дисплея в более жестких условиях расщепления. Указанный процесс позволил выявить вариант I-OnuI (PD-1.ITSM.ex5_RD2_73, SEQ ID NO: 7), который содержал четыре аминокислотные мутации относительно варианта RD1 и имел в несколько раз более высокий процент клеток, экспрессирующих mCherry, по сравнению с вариантом RD1. Фигура 4. Для повышения эффективности редактирования генов на сайте-мишени экзона 5 были выполнены три дополнительных круга скрининга для обогащения активности (PD-1.ITSM.ex5_RD3_03, SEQ ID NO: 8; PD-1.ITSM.ex5_RD4_CV23, SEQ ID NO: 9; и PD-1.ITSM.ex5_RD5_CV23MK, SEQ ID NO: 10).
Пример 2
Характеризация варианта I-OnuI и megaTAL, нацеленных на экзон 5 PD-1
Сайт-мишень экзона 5 PD-1 содержит CpG-динуклеотидные мотивы как в сайте связывания мегануклеазы, так и в соседнем сайте связывания массива TAL (фигура 1 В). Статус метилирования этих динуклеотидов оценивали путем бисульфитного секвенирования в первичных Т-клетках человека, активированных CD3 и CD28 и культивируемых в полной среде с добавкой IL-2. Через 3 дня геномную ДНК выделяли и обрабатывали бисульфитом для превращения любых неметилированных цитозиновых оснований в урацил. Сайт-мишень экзона 5 затем секвенировали для выявления неметилированного (превращенного в тимин) в сравнении с метилированным (оставшимся цитозином) статусом каждого цитозина. Результаты показывают, что оба цитозина с CpG мотивом в сайте-мишени были преимущественно метилированы (Фигура 5), что согласуется с типичными паттернами метилирования «генного тела».
Чтобы подтвердить, что вариант PD-1.ITSM.ex5_RD5_CV23MK хоминг-эндонуклеазы (HE) распознает и расщепляет метилированный сайт-мишень экзона 5, проводили анализ аффинности связывания и расщепления ДНК с использованием субстратов, содержащих 5-метилцитозин в положении p5 сайта-мишени, или обратного комплемента гуанинового основания в p6 или на обеих цепях последовательности-мишени, что является показателем статуса метилирования сайта-мишени в активированных Т-клетках. Фигура 6. Вариант HE связывал и расщеплял метилированные ДНК-субстраты и немодифицированный субстрат аналогичным образом.
Вариант PD-1.ITSM.ex5_RD5_CV23MK HE был отформатирован как megaTAL (SEQ ID NO: 19) путем добавления толерантного к 5-метилцитозину массива TAL величиной 10,5 единиц, который соответствует сайту-мишени массива TAL из 11 пар оснований (SEQ ID NO: 26), к N-концу домена мегануклеазы (как описано Boissel et al., 2013). Последовательность сайта-мишени megaTAL описана в SEQ ID NO: 27. MegaTAL был протестирован на соответствие метилированному динуклеотиду CpG, присутствующему в сайте-мишени массива TAL. Массив TAL был сконструирован так, чтобы быть толерантным к метилированному основанию путем включения «N*» RVD в соответствующую позицию массива.
Эффективность редактирования megaTAL оценивали путем предварительной стимуляции первичных Т-клеток человека анти-CD3 и анти-CD28 антителами в среде с добавлением цитокинов в течение 48-72 часов, а затем подвергали клетки электропорации с транскрибированной in vitro (IVT), кэппированной и полиаденилированной мРНК (SEQ ID NO: 40), кодирующей megaTAL PD-1.ITSM.ex5_RD5_CV23MK. Кроме того, была добавлена IVT-мРНК, кодирующая 3'-5'-экзонуклеазу Trex2, чтобы усилить процессинг разрыва путем негомологичного присоединения конца (NHEJ) (см. Certo et al., 2012). После электропорации клетки культивировали в течение 7-10 дней в среде с цитокинами, в течение которых извлекали аликвоты для выделения геномной ДНК с последующей ПЦР-амплификацией по сайту-мишени экзона 5 PD-1.
Частоту инсерций/делеций измеряли методом отслеживания инсерций/делеций Tracking of Indels DEcomposition (TIDE, см. Brinkman et al., 2014) или высокопроизводительного секвенирования. Эффективность редактирования megaTAL PD-1.ITSM.ex5_RD5_CV23MK в присутствии Trex2 составила приблизительно 60%. Фигура 7А. Преобладающими типами инсерций/делеций, наблюдаемыми на сайте- мишени, были+1, -1, -2, -3 или -4 нуклеотида. Фигура 7B. Указанный анализ подтвердил, что вариант PD-1.ITSM.ex5_RD5_CV23MK разрушил консенсусный мотив ITSM PD-1 в значительной части человеческих T-клеток, обработанных megaTAL.
Пример 3
Перепрограммирование доменов мегануклеазы I-OnuI для разрушения внеклеточных доменов в гене рецептора запрограммированной смерти-1 (pd-1)
Центральная-4-специфичность варианта I-OnuI HE, который нацелен на сайт-мишень экзона 5 PD-1 (SEQ ID NO: 25), была охарактеризована с использованием высокопроизводительных анализов эндонуклеаз in vitro с поверхностным дисплеем дрожжей (Jarjour, West-Foyle et al., 2009). Плазмида, кодирующая вариант HE, который нацелен на экзон 5 PD-1, была трансформирована в S. cerevisiae для поверхностного дисплея, а затем протестирована на активность расщепления в отношении сгенерированных ПЦР двухцепочечных ДНК-субстратов, содержащих последовательность ДНК сайта-мишени экзона 5 PD-1, которая содержит каждую из 256 возможных центральных-4 последовательностей. Полученный профиль специфичности показал, что указанный перепрограммированный I-OnuI является высокоселективным в отношении центральных-4 сайтов-мишеней (Фигура 8), но может также расщеплять дополнительные неканонические центральные-4 сайты-мишени. Поскольку очень мало канонических центральных-4 сайтов-мишеней I-OnuI расположено в других местах гена PD-1, неканонические центральные-4 сайты-мишени в экзоне 1 и экзоне 2 PD-1 были использованы для перепрограммирования дополнительных хоминг-эндонуклеаз.
Эти неканонические центральные-4 сайты-мишени в экзоне 1 и экзоне 2 PD-1 находятся в областях, которые кодируют сигнальный пептид и домен IgV соответственно. Без привязки к какой-либо конкретной теории, предполагается, что нацеливание на эти области путем инделами, редактирующими ген, устраняет или дестабилизирует экспрессию белка PD-1 создавая чистый инактивированный фенотип.Кроме того, имея доступ к этим сайтам для более сложных операций по редактированию гена, включая, помимо прочего, целевую инсерцию трансгенных кассет, можно создавать фенотипы, которые находятся под уникальным регуляторным контролем.
I-OnuI был перепрограммирован так, чтобы быть нацеленным на два неканонических центральных-4 сайта-мишени (SEQ ID NO: 30 и 35), по одному в каждом из этих двух экзонов/мотивов, и распространяющийся на сайты-мишени соответствующего массива TAL (SEQ ID NO: 26 и 31) и сайты-мишени полного megaTAL (SEQ ID NO: 31 и 37). Рис 9А и 9 В. I-OnuI был перепрограммирован так, чтобы быть нацеленным на экзон 1 или экзон 2 гена PD-1 путем конструирования модульных библиотек, содержащих вариабельные аминокислотные остатки в интерфейсе распознавания ДНК. Для конструирования вариантов вырожденные кодоны были встроеены в ДНК-связывающие домены I-OnuI с использованием олигонуклеотидов. Олигонуклеотиды, кодирующие вырожденные кодоны, использовали в качестве ПЦР-матриц для создания библиотек вариантов путем рекомбинации разрывов в штамме дрожжей S. cerevisiae. Каждая библиотека вариантов охватывала N- или С-концевой домен узнавания ДНК I-OnuI и содержала ~ 107-108 уникальных трансформантов. Полученные библиотеки поверхностных дисплеев подвергали скринингу с помощью проточной цитометрии на активность расщепления в отношении сайтов-мишеней, содержащих «полусайты» соответствующих доменов (экзон 1: SEQ ID NO: 34 и 35; экзон 2: SEQ ID NO: 38 и 39).
Дрожжи, имеющие I-OnuI HE с перепрограммированными N- и C-концевыии доменами, очищали и экстрагировали плазмидную ДНК. Проводили реакции ПЦР для амплификации перепрограммированных доменов, которые впоследствии трансформировали в S. cerevisiae для создания библиотек комбинаций перепрограммированных доменов. Выявляли полностью перепрограммированные варианты I-OnuI, активные против полного сайта-мишени в области, кодирующей сигнальный пептид в экзоне 1 PD-1, и в области, кодирующей домен IgV в экзоне 2 PD-1, и выделяли из этих библиотек.
Перепрограммированные I-OnuI HE, нацеленные на сайты-мишени экзона 1 и экзона 2 PD-1, клонировали в плазмиды экспрессии млекопитающих и затем индивидуально трансфицировали в клеточную линию фибробластов HEK 293T, содержащую соответствующие последовательности-мишени в направлении против хода транскрипции от гена вне рамки, кодирующего флуоресцентный белок iRFP.
Перепрограммированный HE I-OnuI, нацеленный на сайт экзона 1 PD-1 (PD-1.ile3.exon1_RD1_B1, SEQ ID NO: 11), показал умеренную эффективность экспрессии iRFP в контексте клеточной хромосомы, либо в виде автономного варианта HE, либо после форматирования в качестве megaTAL (SEQ ID NO: 20). Вторичную библиотеку вариантов I-OnuI генерировали путем проведения случайного мутагенеза по варианту RD1 экзона 1 PD-1, выявленному в процессе скрининга первоначальной библиотеки. Сортировку потока на основе дисплея выполняли в более жестких условиях расщепления, чтобы выделить варианты с улучшенной каталитической эффективностью. Указанный процесс выявил два варианта I-OnuI (PD-1.ile3.exon1_RD2_B1H8, PD-1.ile3.exon1_RD2_B1G2, SEQ ID NO: 12 и 60 соответственно). Эти варианты содержали четыре (PD-1.ile3.exon1_RD2_B1H8) или пять (PD-1.ile3.exon1_RD2_B1G2) аминокислотных мутации относительно варианта RD1 и имели значительно более высокий уровень экспрессирующих iRFP клеток по сравнению с вариантом RD1 экзона 1PD-1, как в качестве автономного варианта HE, так и после форматирования в виде megaTAL (SEQ ID NO: 21 и SEQ ID NO: 64). Фигура 10.
Перепрограммированный HE I-OnuI, нацеленный на сайт экзона 2 PD-1 (PD-1.IgV.exon2_RD1_G5; SEQ ID NO: 13), показал высокую эффективность экспрессии iRFP при доставке либо в виде автономного варианта HE, либо после форматирования в виде megaTAL (SEQ ID NO: 22).
Варианты HE, нацеленные на экзон 1 и экзон 2, проявляли сильные свойства аффинности к ДНК при измерении путем равновесного титрования субстрата с использованием их соответствующих последовательностей-мишеней. Фигура 11A.
Нуклеазу экзона 1 дополнительно очищали для улучшения ее специфичности в области, контактирующей с парой оснований -8, -7 и -6. Микробиблиотеки были построены путем рандомизации аминокислотных остатков 68, 70, 78, 80 и 82, и после 6 раундов сортировки с помощью проточного цитометра три клона (PD-1.ile3.exon1_RD2_B1G2C4, PD-1.ile3.exon1_RD2_B1G2C11 и PD-1.ile3.exon1_RD2_B1G2C5; SEQ ID NO: 61, 62 и 63 соответственно) показали более высокую специфичность, чем родительская нуклеаза (PD-1.ile3.exon1_RD2_B1G2; SEQ ID No: 60). Фигура 11B.
Для оценки статуса метилирования CpG-мотивов, присутствующих в сайтах-мишенях экзона 1 и экзона 2 PD-1, использовали бисульфитное секвенирование. Фигура 12. Было показано, что мотив CpG в сайте-мишени PD-1 экзона 1 в активированных Т-клетках неметилирован, тогда как оба мотива CpG в сайте-мишени экзона 2 PD-1 метилированы. Был выполнен анализ активности на основе дисплея, чтобы подтвердить, что последовательность-мишень с полностью метилированным CpG экзона 2 PD-1 была эффективно расщеплена соответствующим вариантом HE.
Кроме того, сайт-мишень с полностью метилированным CpG экзона 2 был использован для выявления варианта I-OnuI (PD-1.IgV.exon2_RD1_PS3, SEQ ID NO: 14) с улучшенной связывающей и расщепляющей активностью в отношении CpG- метилированного сайта-мишени.
Пример 4
Направленное разрушение гена pd-1 в первичных человеческих т-клетках спасает pd-l1-опосредованное подавление функции т-клеток
Функциональное воздействие megaTAL, перепрограммированных для расщепления различных последовательностей-мишеней в гене PD-1, оценивали в первичных Т-клетках человека, активированных с помощью CD3 и CD28 и культивируемых в полной среде с добавлением IL-2. Активированные PBMC трансдуцировали лентивирусным вектором, кодирующим анти-BCMA CAR. Т-клетки с анти-ВСМА CAR подвергали электропорации транскрибированной in vitro мРНК, кодирующей megaTAL, нацеленные либо на экзон 5 PD-1, либо на экзон 1 PD-1 (SEQ ID NO: 40 и 41, соответственно), и мРНК, кодирующей Trex2 (SEQ ID NO: 43). Контрольные клетки включали необработанные Т-клетки или Т-клетки, обработанные мРНК, кодирующей голубой флуоресцентный белок (CFP) или megaTAL, нацеленный на CCR5 (см. Sather et. al., Sci Transl Med. 2015 Sep 30;7(307):307ra156). После 10-дневной экспансии Т-клетки стимулировали поликлональным активирующим реагентом форбол-12-миристат-13-ацетат (PMA)/иономицин (P/I). PD-1 естественным образом активируется на клеточной поверхности после активации Т-клеток. Активация PD-1 была подавлена после трансфекции мРНК megaTAL экзона 1 PD-1, что указывает на то, что инделы в данной области нарушает нормальную выработку белка PD-1. Напротив, обработка мРНК, кодирующей контрольный CCR5-megaTAL или нацеленным на экзон 5 PD-1, megaTAL, не оказала влияния на поверхностную экспрессию PD-1, несмотря на высокий уровень инделов, индуцированных в ITSM этим megaTAL. Фигура 13А. Дальнейшие эксперименты, повторенные в сходных условиях с обогащенными по специфичности версиями мРНК экзона 1 PD-1 (SEQ ID NO: 65, 66, 67 и 68), также показали сходное нарушение экспрессии PD-1 в Т-клетках. Фигура 13B
Одновременная доставка megaTAL экзона 1 и экзона 5 PD-1 значительно улучшала нарушение экспрессии PD-1 на клеточной поверхности, независимо от доставки экзонуклеазы Trex2. Фигура 14. Это указывает на то, что одновременное образование проксимального разрыва ДНК является механизмом, способствующим событиям делеции большого гена с высокой эффективностью, не зависящей от экзонуклеазного усиления индела.
Влияние нарушения передачи сигналов PD-1 в Т-клетках путем нацеливания либо на его экспрессию, либо на его сигнальные функции анализировали на основании выработки цитокинов в клетке CAR-T в ответ на опухолевые клетки, модифицированные для экспрессии лиганда PD-1, PD-L1. Совместное культивирование анти-BCMA CAR T-клеток с BCMA-экспрессирующими опухолевыми клеточными линиями приводило к активации T-клеток и последующей секреции воспалительных цитокинов, примером чего являются высокие уровни TNFα и IL-17A, измеренные в супернатанте. Коэкспрессия PD-L1 на BCMA-экспрессирующих опухолевых клетках подавляла выработку воспалительных цитокинов. Однако трансфекция анти-BCMA CAR T-клеток мРНК, кодирующей megaTALs либо экзона 1 либо экзона 5 PD-1, снижает опосредованную PD-L1 супрессию цитокинов, так как выработка воспалительных цитокинов в этих образцах восстанавливается до базовых уровней Фигура 15A.
Пример 5
Гомологичная рекомбинация трансгена в экзон 1 гена PD-1
Была разработана и сконструирована плазмида рекомбинантного адено-ассоциированного вируса (rAAV), содержащая промотор-трансгенную кассету, содержащую гетерологичный промотор, трансген, кодирующий флуоресцентный белок, и сигнал полиаденилирования, расположенный между областями гомологии, нацеленными на гены. Целостность элементов AAV ITR была проверена с помощью лизата XmaI. Трансгенную кассету помещали между двумя гомологичными областями, длиной приблизительно 600-700 п.н., фланкирующими сайт расщепления megaTAL экзона 1 PD-1 (SEQ ID NO: 27). 5'-гомологичное плечо (SEQ ID NO: 54) содержало часть первого экзона PD-1 и другие последовательности в направлении против транскрипции от сайта расщепления megaTAL. 3'-гомологичное плечо (SEQ ID NO: 55) содержало часть экзона PD-1 и другие последовательности, расположенные по ходу транскрипции от сайта расщепления megaTAL. Ни одна гомологичная область не содержала полного сайта-мишени megaTAL. Указанная иллюстративная кассета экспрессии содержала промотор энхансера вируса миелопролиферативной саркомы с делецией опосредующего отрицательную регуляцию участка, с заменой на праймер-связывающий сайт из dl587rev (MND), функционально связанный с полинуклеотидом, кодирующим флуоресцентный полипептид, например синий флуоресцентный белок (BFP), красный флуоресцентный белок (RFP), голубой флуоресцентный белок (CFP), зеленый флуоресцентный белок (GFP) и т.д. Кроме того, кассета экспрессии содержала сигнал позднего полиаденилирования SV40, расположенный по ходу транскрипции от кодона терминации трансгена. Фигура 16А. Рекомбинантный AAV (rAAV) получали путем транзиторной котрансфекции клеток HEK 293T плазмидами, обеспечивающими репликационные, капсидные и аденовирусные хелперные элементы, необходимые для выработки вируса. Рекомбинантный rAAV был выделен из совместно трансфицированной культуры клеток HEK 293T с использованием ультрацентрифугирования в градиенте йодиксанола.
MegaTAL-индуцированную гомологичную рекомбинацию оценивали в первичных Т-клетках человека, активированных CD3 и CD28 и культивируемых в полной среде с добавлением IL-2. После 3 дней культивирования Т-клетки промывали и подвергали электропорации транскрибированной in vitro мРНК megaTAL экзона 1 PD-1 (SEQ ID NO: 41), а затем трансдуцировали очищенной трансгенной кассетой, кодирующей рекомбинантный AAV, MND-GFP (SEQ ID NO: 53). Контрольная группа включала Т-клетки, обработанные мРНК megaTAL или вектором, нацеленным на rAAV. Использовали проточную цитометрию для измерения в нескольких временных точках доли Т-клеток, экспрессирующих флуоресцентный белок, и для дифференциации транзитороной экспрессии флуоресцентного белка из эписомального rAAV-нацеленного вектора от долговременной экспрессии хромосомно интегрированной кассеты. Фигура 16B. Опосредованное MegaTAL разрушение гена PD-1 обнаруживали секвенированием и по потере экспрессии PD-1 после активации поликлональных Т-клеток.
Долговременную экспрессию трансгена наблюдали в 20-60% Т-клеток, которые были обработаны вектором, нацеленным как на megaTAL, так и на rAAV. В контрольных образцах обработка только rAAV приводила к вариабельным уровням транзитороной экспрессии флуоресцентного белка и очень низким уровням (<1%) длительной экспрессии флуоресцентного белка в обработанных Т-клетках, что согласуется с отсутствием интеграции в геном. Результаты были подтверждены в экспериментах, проведенных на Т-клетках, выделенных от нескольких независимых доноров.
Пример 6
Гомологичная рекомбинация трансгена, кодирующего химерный антигенный рецептор (CAR) в ген PD-1
Плазмиды рекомбинантного адено-ассоциированного вируса (rAAV) разрабатывали, конструировали и верифицировали, как описано выше, за исключением того, что трансгенная кассета, кодирующая анти-CD19 CAR (SEQ ID NO: 56) или анти-BCMA CAR (SEQ ID NO: 57) была помещена между гомологичными областями направленным на экзон 1 PD-1. Кассеты экспрессии CAR содержали промотор MND, функционально связанный с полинуклеотидом, кодирующим CAR, содержащим полученный из CD8α сигнальный пептид, одноцепочечный вариабельный фрагмент (scFv), нацеленный либо на антиген CD19, либо на антиген созревания В-клеток (BCMA), полученная из CD8α. шарнирная область и трансмембранный домен, внутриклеточный костимулирующий домен 4-1BB, CD3 дзета-сигнальный домен и гомологичные плечи, предназначенные для нацеливания на сайт-мишень экзона 1 PDCD1 (SEQ ID NO: 54 и 55).
Первичные человеческие Т-клетки активировали CD3 и CD28 и выращивали в среде с добавками цитокинов, как описано выше. Гомологичную рекомбинацию трансгенов CAR в экзон 1 PD-1 сайт-мишень оценивали с использованием активированных первичных человеческих Т-клеток, электропорированных in vitro транскрибированной мРНК экзона 1 PD-1 (SEQ ID NO: 41), а затем трансдуцированных кодирующим rAAV анти-CD19 или анти-ВСМА CAR. Проточную цитометрию для определения экспрессии CAR проводили через 10 дней, то есть через 7 дней после электропорации и трансдукции. Контрольные образцы включали Т-клетки, обработанные либо мРНК megaTAL, либо вектором, нацеленным на rAAV. Экспрессию CD19-CAR и BCMA-CAR анализировали с использованием рекомбинантных PE-конъюгированных CD19-Fc или PE-конъюгированных BCMA-Fc окрашивающих реагентов. Т-клетки, обработанные мРНК megaTAL и rAAV-CAR, показали экспрессию CD19-CAR в 30-60% Т-клеток и экспрессию BCMA-CAR в 10-20% Т-клеток. Фигура 16B. Наблюдались сходные скорости размножения Т-клеток и неразличимые фенотипы Т-клеток между необработанными, rAAV-обработанными и megaTAL/rAAV CAR-обработанными Т-клетками.
Пример 7
Гомологичная рекомбинация лишенного промотора трансгена в ген pd-1
Была разработана, сконструирована и верифицирована плазмида рекомбинантного адено-ассоциированного вируса (rAAV), содержащая флуоресцентный репортерый трансген (mCherry) и сигнал полиаденилирования, но без экзогенного промотора (SEQ ID NO: 58). Фигура 17. Стартовый кодон mCherry сливается и перекрывается с эндогенным стартовым кодоном PD-1, заменяя остальную часть экзона 1 PD-1 на кДНК, кодирующую белок mCherry. Эта стратегия стимулирует экспрессию флуоресцентного белка от эндогенного промотора PD-1, а также нарушает нормальную экспрессию белка PD-1.
Первичные человеческие Т-клетки активировали, электропорировали транскрибированной мРНК megaTAL in vitro и трансдуцировали rAAV, как описано выше. Контрольные клетки включали Т-клетки, обработанные мРНК megaTAL или вектором, нацеленным на rAAV. Экспрессию флуоресцентного репортера анализировали с помощью проточной цитометрии в различные моменты времени после трансфекции, в присутствии или в отсутствие активации поликлональных Т-клеток, используя форбол-12-миристат-13-ацетат (PMA)/иономицин (P/I). В Т-клетках, обработанных только мРНК megaTAL или вектором, нацеленным на rAAV, экспрессия репортера не наблюдалась. Подобные показатели активности megaTAL наблюдались с или без трансдукции rAAV. Низкоуровневая экспрессия флуоресцентного репортера наблюдалась в Т-клетках, которые получали как megaTAL, так и нацеливающий на rAAV вектор, и были активированы P/I в течение 48 часов. Экспрессия флуоресцентного репортера, обусловленная эндогенным промотором PD-1, была ниже по сравнению с экспрессией рецептора, управляемого гетерологичным промотором (снижение интенсивности флуоресценции в ~5 раз).
Пример 8
Конститутивная экспрессия PD-1 после гомологичной рекомбинации трансгена, кодирующего флуоресцентный белок, в экзон 5 гена PD-1
Была разработана, сконструирована и верифицирована рекомбинантная плазмида адено-ассоциированного вируса (rAAV), содержащая гетерологичный промотор, трансген синего флуоресцентного белка (BFP) и сигнал полиаденилирования (SEQ ID NO: 44) для введения в экзон 5 PD-1. Трансгенная кассета была помещена между 1,3 т.п.н. и 1,0 т.п.н.гомологичными плечами, фланкирующими сайт расщепления megaTAL экзона 5 PD-1. 5'-гомологичное плечо (SEQ ID NO: 45) включало в себя часть экзона 5 PD-1 в направлении против хода транскрипции от сайта megaTAL, и другие последовательности в направлении против хода транскрипции. 3'-гомологичное плечо (SEQ ID NO: 46) содержало концевую кодирующую последовательность экзона 5 PD-1 и другие последовательности по ходу транскрипции. Ни одна гомологичная область не содержала полного сайта-мишени megaTAL. Фигура 18.
Первичные человеческие Т-клетки активировали CD3 и CD28, электропорировали in vitro транскрибированной мРНК megaTAL экзона 5 PD-1 (SEQ ID NO: 40), затем трансдуцировали rAAV, кодирующим трансген флуоресцентного репортера. Контрольные клетки включали необработанные Т-клетки и Т-клетки, обработанные только вектором, нацеленным на rAAV. Экспрессию флуоресцентного репортера анализировали проточной цитометрией в различные моменты времени после трансфекции. Клетки, обработанные одним rAAV, демонстрировали транзиторную экспрессию BFP, которая снижалась до фоновых уровней во время размножения клеток. Т-клетки, которые получали как мРНК megaTAL экзона 5 PD-1, так и вектор rAAV, демонстрировали стабильную популяцию Т-клеток, экспрессирующих BFP, которые сохранялись в течение всего периода культивирования ex vivo.
Т-клетки, обработанные мРНК megaTAL экзона 5 PD-1 и вектором rAAV, имели отчетливый паттерн экспрессии PD-1, в результате чего в отсутствие активации почти все BFP-экспрессирующие клетки также экспрессировали белок PD-1. Это указывает на то, что стратегия нацеливания изменила нормальную регуляцию гена PD-1 на конститутивный, а не индуцибельный паттерн экспрессии. Более того, благодаря нацеливанию на экзон 5 PD-1 конститутивно экспрессируемый белок в этой стратегии нацеливания представляет собой вариант PD-1, который обладает нарушенной ингибирующей передачей сигналов и который может действовать как доминантный отрицательный рецептор. Экспрессия PD-1 не была повышена в Т-клетках, обработанных мРНК 5 megaTAL экзона PD-1 или в Т-клетках, обработанных мРНК megaTAL и rAAV, содержащих гомологичные плечи к экзону 1. Фигура 19.
Пример 9
Гомологичная рекомбинация трансгена в ген PD-1 не зависит от одиночного нуклеотидного полиморфизма (SNP)
Локус PD-1 является генетически гетерогенным, с высокой распространенностью SNP, присутствующих как в интронных, так и в экзонных областях. Присутствие дивергентных SNP вблизи сайта расщепления megaTAL может потенциально влиять на эффективность гомологичной рекомбинации в локусе PD-1. Плазмиды рекомбинантного адено-ассоциированного вируса (rAAV), включающие гетерологичный промотор, трансген флуоресцентного белка и поздний сигнал полиаденилирования SV40, были разработаны для оценки воздействия SNP, затем сконструированы и верифицированы, как описано выше (SEQ ID NO: 47 и 48). Гомологичные плечи были либо идентичны консенсусной последовательности PD-1, либо они были разработаны для включения точечных мутаций из обычных SNP, проксимальных к сайту нацеливания на megaTAL экзона 5 PD-1. В этих конструкциях 5'-гомологичное плечо (L-SNP, SEQ ID NO: 49) содержит интронный G/A SNP (rs6705653), расположенный в 220 п.н. в направлении против хода транскрипции от сайта расщепления megaTAL, в то время как 3'-гомологичное плечо (R-SNP, SEQ ID NO: 50) содержит молчащий C/T SNP (rs2227981), расположенный в 68 п.н. по ходу транскрипции от сайта расщепления megaTAL PD-1. Фигура 20.
Первичные человеческие Т-клетки активировали CD3 и CD28, электропорировали транскрибированной in vitro мРНК megaTAL и трансдуцировали rAAV, кодирующим консенсусные гомологичные плечи, или rAAV, несущим либо L-SNP, либо R-SNP гомологичные плечи. Контрольные клетки включали Т-клетки, трансдуцированные только направленным вектором rAAV. Экспрессию флуоресцентного белка анализировали проточной цитометрией в разные моменты времени после трансдукции. Экспрессия флуоресцентного белка снизилась до фоновых уровней в образцах, которые получали только rAAV, в то время как Т-клетки, обработанные megaTAL в сочетании с rAAV, продемонстрировали стабильные уровни экспрессии флуоресцентного белка. Все образцы демонстрировали схожие уровни флуоресценции, несмотря на то, что их обрабатывали либо не-SNP-содержащими rAAV векторами, либо векторами, содержащими L -или R-варианты SNP, что указывает на то, что SNP, проксимальные к сайту-мишени megaTAL, не влияют или не оказывают существенного влияния на HR на сайте-мишени.
Пример 10
Гомологичная рекомбинация трансгена, кодирующего рецептор-переключатель, в локус PDCD1
Рецепторы-переключатели представляют собой сконструированные химерные молекулы, способные преобразовывать внеклеточные ингибирующие сигналы в сигналы внутриклеточной активации, однако их эффективность часто коррелирует с их способностью подавлять нативную экспрессию природных рецепторов. Один из способов обойти это ограничение - встроить рецептор-переключатель в нативный локус и нарушить естественный рецептор. Плазмида рекомбинантного адено-ассоциированного вируса (rAAV), содержащая гетерологичный промотор, трансген, кодирующий внеклеточный лиганд-связывающий домен PD-1 и внутриклеточный сигнальный домен CD28 (рецептор-переключатель PD-1, SEQ ID NO: 59), и поздний сигнал полиаденилирования SV40, была разработана для нацеливания на экзон 5 PD-1, сконструирована и верифицирована. 5'-гомологичное плечо включало в себя часть экзона 5 PD-1 в направлении против хода транскрипции от сайта расщепления megaTAL, содержащего ITIM и другие последовательности в направлении против хода транскрипции. 3'-гомологичное плечо содержало концевую кодирующую последовательность экзона 5 PD-1 и часть UTR-области и было укорочено до ~ 650 п.н. для размещения кДНК рецептора-переключателя. Ни одна гомологичная область не содержала полного сайта-мишени megaTAL. Фигура 21.
Первичные Т-клетки человека активировали CD3 и CD28, электропорировали in vitro транскрибированной мРНК экзона 5 megaTAL экзона PD-1 и трансдуцировали направленным на rAAV вектором, кодирующим рецептор-переключатель PD-1. Контрольные клетки включали образцы, которые получали только megaTAL или rAAV. Экспрессию рецептора-переключателя PD-1 анализировали путем окрашивания анти-PD-1 антителом в отсутствие стимуляции Т-клеток. Наблюдались высокие уровни экспрессии в Т-клетках, обработанных мРНК megaTAL экзона 5 PD-1 и трансдуцированных rAAV.
Пример 11
Дальнодействующее вырезание внутриклеточной сигнальной области PD-1 с применением HDR
Известно, что механизм гомологичной рекомбинации включает механизмы «поиска» гомологии, которые могут охватывать отдаленные геномные последовательности. Без привязки к какой-либо конкретной теории, предполагается, что более существенные участки генов-мишеней или хромосомных областей могут быть подвержены эффективному манипулированию с применением комбинации доставки megaTAL и rAAV. Была разработана, сконструирована и верифицирована плазмида рекомбинантного адено-ассоциированного вируса (rAAV), включающая гетерологичный промотор, трансген BFP и поздний сигнал полиаденилирования SV40, фланкированный гомологичными плечами для оценки дистальных событий HR (SEQ ID NO: 52). Трансген был фланкирован 5'-гомологичным плечом 1,3 т.п.н., начинающимся направлении против хода транскрипции от экзона 3 PD-1 и заканчивающимся непосредственно перед началом экзона 4. 3'- гомологичное плечо содержало концевую кодирующую последовательность экзона 5 PD-1 и часть UTR области. Ни одна гомологичная область не содержала полного сайта-мишени megaTAL. Успешная гомологичная рекомбинация предназначена для устранения всей внутриклеточной сигнальной области PD-1, чтобы при этом на обработанной клетке оставалась только внеклеточная и трансмембранная часть PD-1. Фигура 22.
Первичные человеческие Т-клетки активируют CD3 и CD28, электропорируют транскрибируемой in vitro мРНК megaTAL и трансдуцируют BFP, кодирующим нацеленный на rAAV вектор, содержащий дистальне 5'-гомологичное плечо. Контрольные клетки включают Т-клетки, обработанные вектором, нацеленным на megaTAL или rAAV. Стабильная экспрессия BFP наблюдается только в образцах, которые получили мРНК megaTAL, нацеленного на экзон 5 и rAAV, и PDCD1.
Пример 12
Разрушение гена PD-1 и антитело-опосредованная блокада PD1
в первичных Т-клетках человека in vitro
Функциональное воздействие megaTAL, перепрограммированных для расщепления различных последовательностей-мишеней в гене PD-1, оценивали в первичных Т-клетках человека, активированных CD3 и CD28 и культивируемых в полной среде с добавлением IL-2. Активированные PBMC трансдуцировали лентивирусным вектором, кодирующим анти-BCMA CAR. Анти-BCMA CAR-Т-клетки подвергали электропорации транскрибированной in vitro мРНК, кодирующей megaTAL, нацеленные либо на экзон 5 PD-1, либо на экзон 1 PD-1 (SEQ ID NO: 40 и 41, соответственно), и мРНК, кодирующей Trex2 (SEQ ID NO: 43). Контрольные клетки включали Т-клетки, электропорированные без мРНК (имитационные Mock EP), или Т-клетки, электропорированные мРНК, кодирующей megaTAL, специфичный для TCRα, который не обладает каталитической активностью.
После 10-дневного размножения Т-клетки совместно культивировали с клетками А549, трансдуцированными лентивирусным вектором, кодирующим BCMA-GFP. CAR Т-клетки совместно культивировали с клетками-мишенями A549 отдельно или в присутствии 20 мкг/мл анти-PD-1-антитела. Клетки совместно культивировали в течение 72 часов, и анализировали уровни цитокинов в супернатантах с применением анализа с использованием микросфер (Intellicyt QBeads). В отсутствие анти-PD-1-антитела клетки, которые были обработаны megaTAL либо PD1-Ex1, либо PD1-Ex5, показали повышенную выработку IFNγ и TNFα по сравнению с контрольными клетками - имитационными Mock EP и TCRα. Добавление антитела αPD-1 устраняет эту разницу, что приводит к эквивалентному уровню секреции IFNγ и TNFα в супернатантах, полученных из контрольных и обработанных PD1 megaTAL клеток (Фигура 15B).
Пример 13
Гомологичная рекомбинация трансгена, лишенного промотора, в ген PD-1
Была разработана, сконструирована и верифицирована плазмида рекомбинантного адено-ассоциированного вируса (rAAV), содержащая трансген флуоресцентного репортера (mCherry) и сигнал полиаденилирования, но без экзогенного промотора (SEQ ID NO: 58). Фигура 17. Стартовый кодон mCherry сливается и перекрывается с эндогенным стартовым кодоном PD-1, при этом заменяя остальную часть экзона 1 PD-1 на кДНК, кодирующую белок mCherry. Эта стратегия стимулирует экспрессию флуоресцентного белка из эндогенного промотора PD-1, а также нарушает нормальную экспрессию белка PD-1.
Первичные человеческие Т-клетки активировали, электропорировали транскрибированной in vitro мРНК megaTAL и трансдуцировали rAAV, как описано выше. Контрольные образцы включали Т-клетки, обработанные мРНК megaTAL или вектором, нацеленным на rAAV. Экспрессию флуоресцентного репортера анализировали с помощью проточной цитометрии через 24 часа после стимуляции в присутствии или в отсутствие активации поликлональных Т-клеток с использованием форбол-12-миристат-13-ацетата (PMA)/иономицина (P/I). В необработанных Т-клетках экспрессия megaTAL не наблюдалась. Перед стимуляцией P/I низкие базальные уровни экспрессии флуоресцентного репортера наблюдались в Т-клетках, которые получали и megaTAL и нацеленный на rAAV вектор для HDR. Двадцать четыре часа стимуляции P/I увеличивали экспрессию флуоресцентного репортера, вызванную эндогенным PD-1, с 3% до 27,3% (сравните верхнюю и нижнюю правые панели на рисунке 17).
Пример 14
Гомологичная рекомбинация цитокинового трансгена, лишенного промотора, в локус PD-1
Была разработана, сконструирована и верифицирована плазмида рекомбинантного адено-ассоциированного вируса (rAAV), содержащая трансгены IL-12 или IL-15 и сигнал полиаденилирования, но без экзогенного промотора (SEQ ID NO: 58). Стартовый кодон трансгена сливается и перекрывается с эндогенным стартовым кодоном PD-1, заменяя остальную часть экзона 1 PD-1 на кДНК, кодирующую цитокин IL-12 или IL-15 (Фигура 23, верхняя панель). Эта стратегия стимулирует экспрессию трансгена из эндогенного промотора PD-1, а также нарушает нормальную экспрессию PD-1.
Первичные человеческие Т-клетки активировали и трансдуцировали лентивирусным вектором, кодирующим анти-BCMA CAR, через 24 часа после активации. Клетки размножали в течение 2 дней, электропорировали транскрибированной in vitro megaTAL мРНК и трансдуцировали rAAV, как описано выше. Контрольные образцы включали нетрансдуцированные T-клетки, T-клетки, обработанные только анти-BCMA CAR, или T-клетки, обработанные анти-BCMA CAR и культивированные в присутствии рекомбинантного IL-12. Активность megaTAL PD-1 определяли с помощью анализа проточной цитометрии экспрессии PD-1 на T-клетках, стимулированных в течение 24 часов PMA/иономицином. Сочетание megaTAL PD-1 с IL-12 AAV показало снижение экспрессии PD-1 приблизительно на 40% по сравнению с контрольными образцами (Фигура 23, нижняя панель).
Уровни экспрессии IL-12 измеряли с помощью ELISA после активации PMA/иономицином или после совместного культивирования Т-клеток с экспрессирующей антиген клеточной линией K562-BCMA. Нестимулированные T-клетки показали минимальную выработку IL-12, а стимуляция PMA/иономицином при совместном культивировании с антиген-позитивными клетками K562 приводила к секреции IL-12 (фигура 24A). Анализы повторной стимуляции использовали для измерения функционального истощения BCMA-CAR Т-клеток. Опухолевые клетки и анти-BCMA CAR Т-клетки смешивали, культивировали в течение 7 дней и добавляли дополнительные клетки-мишени K562-BCMA для имитации повторной стимуляции. После 1-й стимуляции уровни выработки IFNγ и цитотоксичности были одинаковыми во всех образцах Т-клеток. После 2-й стимуляции выработка IFNγ и цитотоксичность увеличились в анти-BCMA-CAR T-клетках, обработанных рекомбинантным IL-12 или обработанных и megaTAL PD-1, и HDR матрицей IL-12, по сравнению с контрольными обработанными клетками (фигура 24B).
В общем, в следующей формуле изобретения используемые термины не должны толковаться как ограничивающие формулу изобретения конкретными вариантами реализации, раскрытыми в описании и формуле изобретения, но должны толковаться как включающие в себя все возможные варианты реализации вместе с полным объемом эквивалентов, подразумеваемых такими пунктами формулы. Соответственно, формула изобретения не ограничивается приведенным описанием.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110>БЛУБЕРД БИО, ИНК.
МАНН, Джасдип
ГАЙ, Джоэл
ДЖАРДЖУР, Джордан
ЧЖАН, Джой
<120>ВАРИАНТЫ, КОМПОЗИЦИИ И МЕТОДЫ ИСПОЛЬЗОВАНИЯ ХОМИНГ-ЭНДОНУКЛЕАЗЫ PD-1
<130>BLBD-076/02WO
<150>США 62/414 279
<151>2016-10-28
<150>США 62/385 079
<151>2016-09-08
<160>123
<170>PatentIn версия 3.5
<210>1
<211>303
<212>БЕЛОК
<213>Ophiostoma novo-ulmi subsp.americana (митохондрия)
<400>1
Met Ala Tyr Met Ser Arg Arg Glu Ser Ile Asn Pro Trp Ile Leu Thr
1 5 10 15
Gly Phe Ala Asp Ala Glu Gly Ser Phe Leu Leu Arg Ile Arg Asn Asn
20 25 30
Asn Lys Ser Ser Val Gly Tyr Ser Thr Glu Leu Gly Phe Gln Ile Thr
35 40 45
Leu His Asn Lys Asp Lys Ser Ile Leu Glu Asn Ile Gln Ser Thr Trp
50 55 60
Lys Val Gly Val Ile Ala Asn Ser Gly Asp Asn Ala Val Ser Leu Lys
65 70 75 80
Val Thr Arg Phe Glu Asp Leu Lys Val Ile Ile Asp His Phe Glu Lys
85 90 95
Tyr Pro Leu Ile Thr Gln Lys Leu Gly Asp Tyr Met Leu Phe Lys Gln
100 105 110
Ala Phe Cys Val Met Glu Asn Lys Glu His Leu Lys Ile Asn Gly Ile
115 120 125
Lys Glu Leu Val Arg Ile Lys Ala Lys Leu Asn Trp Gly Leu Thr Asp
130 135 140
Glu Leu Lys Lys Ala Phe Pro Glu Ile Ile Ser Lys Glu Arg Ser Leu
145 150 155 160
Ile Asn Lys Asn Ile Pro Asn Phe Lys Trp Leu Ala Gly Phe Thr Ser
165 170 175
Gly Glu Gly Cys Phe Phe Val Asn Leu Ile Lys Ser Lys Ser Lys Leu
180 185 190
Gly Val Gln Val Gln Leu Val Phe Ser Ile Thr Gln His Ile Lys Asp
195 200 205
Lys Asn Leu Met Asn Ser Leu Ile Thr Tyr Leu Gly Cys Gly Tyr Ile
210 215 220
Lys Glu Lys Asn Lys Ser Glu Phe Ser Trp Leu Asp Phe Val Val Thr
225 230 235 240
Lys Phe Ser Asp Ile Asn Asp Lys Ile Ile Pro Val Phe Gln Glu Asn
245 250 255
Thr Leu Ile Gly Val Lys Leu Glu Asp Phe Glu Asp Trp Cys Lys Val
260 265 270
Ala Lys Leu Ile Glu Glu Lys Lys His Leu Thr Glu Ser Gly Leu Asp
275 280 285
Glu Ile Lys Lys Ile Lys Leu Asn Met Asn Lys Gly Arg Val Phe
290 295 300
<210>2
<211>303
<212>БЕЛОК
<213>Ophiostoma novo-ulmi subsp.americana (митохондрия)
<400>2
Met Ala Tyr Met Ser Arg Arg Glu Ser Ile Asn Pro Trp Ile Leu Thr
1 5 10 15
Gly Phe Ala Asp Ala Glu Gly Ser Phe Leu Leu Arg Ile Arg Asn Asn
20 25 30
Asn Lys Ser Ser Val Gly Tyr Ser Thr Glu Leu Gly Phe Gln Ile Thr
35 40 45
Leu His Asn Lys Asp Lys Ser Ile Leu Glu Asn Ile Gln Ser Thr Trp
50 55 60
Lys Val Gly Val Ile Ala Asn Ser Gly Asp Asn Ala Val Ser Leu Lys
65 70 75 80
Val Thr Arg Phe Glu Asp Leu Lys Val Ile Ile Asp His Phe Glu Lys
85 90 95
Tyr Pro Leu Ile Thr Gln Lys Leu Gly Asp Tyr Lys Leu Phe Lys Gln
100 105 110
Ala Phe Ser Val Met Glu Asn Lys Glu His Leu Lys Glu Asn Gly Ile
115 120 125
Lys Glu Leu Val Arg Ile Lys Ala Lys Leu Asn Trp Gly Leu Thr Asp
130 135 140
Glu Leu Lys Lys Ala Phe Pro Glu Asn Ile Ser Lys Glu Arg Ser Leu
145 150 155 160
Ile Asn Lys Asn Ile Pro Asn Phe Lys Trp Leu Ala Gly Phe Thr Ser
165 170 175
Gly Glu Gly Cys Phe Phe Val Asn Leu Ile Lys Ser Lys Ser Lys Leu
180 185 190
Gly Val Gln Val Gln Leu Val Phe Ser Ile Thr Gln His Ile Lys Asp
195 200 205
Lys Asn Leu Met Asn Ser Leu Ile Thr Tyr Leu Gly Cys Gly Tyr Ile
210 215 220
Lys Glu Lys Asn Lys Ser Glu Phe Ser Trp Leu Asp Phe Val Val Thr
225 230 235 240
Lys Phe Ser Asp Ile Asn Asp Lys Ile Ile Pro Val Phe Gln Glu Asn
245 250 255
Thr Leu Ile Gly Val Lys Leu Glu Asp Phe Glu Asp Trp Cys Lys Val
260 265 270
Ala Lys Leu Ile Glu Glu Lys Lys His Leu Thr Glu Ser Gly Leu Asp
275 280 285
Glu Ile Lys Lys Ile Lys Leu Asn Met Asn Lys Gly Arg Val Phe
290 295 300
<210>3
<211>303
<212>БЕЛОК
<213>Ophiostoma novo-ulmi subsp.americana (митохондрия)
<220>
<221>MOD_RES
<222>(1)..(3)
<223>Любая аминокислота или отсутствует
<400>3
Xaa Xaa Xaa Met Ser Arg Arg Glu Ser Ile Asn Pro Trp Ile Leu Thr
1 5 10 15
Gly Phe Ala Asp Ala Glu Gly Ser Phe Leu Leu Arg Ile Arg Asn Asn
20 25 30
Asn Lys Ser Ser Val Gly Tyr Ser Thr Glu Leu Gly Phe Gln Ile Thr
35 40 45
Leu His Asn Lys Asp Lys Ser Ile Leu Glu Asn Ile Gln Ser Thr Trp
50 55 60
Lys Val Gly Val Ile Ala Asn Ser Gly Asp Asn Ala Val Ser Leu Lys
65 70 75 80
Val Thr Arg Phe Glu Asp Leu Lys Val Ile Ile Asp His Phe Glu Lys
85 90 95
Tyr Pro Leu Ile Thr Gln Lys Leu Gly Asp Tyr Lys Leu Phe Lys Gln
100 105 110
Ala Phe Ser Val Met Glu Asn Lys Glu His Leu Lys Glu Asn Gly Ile
115 120 125
Lys Glu Leu Val Arg Ile Lys Ala Lys Leu Asn Trp Gly Leu Thr Asp
130 135 140
Glu Leu Lys Lys Ala Phe Pro Glu Asn Ile Ser Lys Glu Arg Ser Leu
145 150 155 160
Ile Asn Lys Asn Ile Pro Asn Phe Lys Trp Leu Ala Gly Phe Thr Ser
165 170 175
Gly Glu Gly Cys Phe Phe Val Asn Leu Ile Lys Ser Lys Ser Lys Leu
180 185 190
Gly Val Gln Val Gln Leu Val Phe Ser Ile Thr Gln His Ile Lys Asp
195 200 205
Lys Asn Leu Met Asn Ser Leu Ile Thr Tyr Leu Gly Cys Gly Tyr Ile
210 215 220
Lys Glu Lys Asn Lys Ser Glu Phe Ser Trp Leu Asp Phe Val Val Thr
225 230 235 240
Lys Phe Ser Asp Ile Asn Asp Lys Ile Ile Pro Val Phe Gln Glu Asn
245 250 255
Thr Leu Ile Gly Val Lys Leu Glu Asp Phe Glu Asp Trp Cys Lys Val
260 265 270
Ala Lys Leu Ile Glu Glu Lys Lys His Leu Thr Glu Ser Gly Leu Asp
275 280 285
Glu Ile Lys Lys Ile Lys Leu Asn Met Asn Lys Gly Arg Val Phe
290 295 300
<210>4
<211>303
<212>БЕЛОК
<213>Ophiostoma novo-ulmi subsp.americana (митохондрия)
<220>
<221>MOD_RES
<222>(1)..(4)
<223>Любая аминокислота или отсутствует
<220>
<221>MOD_RES
<222>(302)..(303)
<223>Любая аминокислота или отсутствует
<400>4
Xaa Xaa Xaa Xaa Ser Arg Arg Glu Ser Ile Asn Pro Trp Ile Leu Thr
1 5 10 15
Gly Phe Ala Asp Ala Glu Gly Ser Phe Leu Leu Arg Ile Arg Asn Asn
20 25 30
Asn Lys Ser Ser Val Gly Tyr Ser Thr Glu Leu Gly Phe Gln Ile Thr
35 40 45
Leu His Asn Lys Asp Lys Ser Ile Leu Glu Asn Ile Gln Ser Thr Trp
50 55 60
Lys Val Gly Val Ile Ala Asn Ser Gly Asp Asn Ala Val Ser Leu Lys
65 70 75 80
Val Thr Arg Phe Glu Asp Leu Lys Val Ile Ile Asp His Phe Glu Lys
85 90 95
Tyr Pro Leu Ile Thr Gln Lys Leu Gly Asp Tyr Lys Leu Phe Lys Gln
100 105 110
Ala Phe Ser Val Met Glu Asn Lys Glu His Leu Lys Glu Asn Gly Ile
115 120 125
Lys Glu Leu Val Arg Ile Lys Ala Lys Leu Asn Trp Gly Leu Thr Asp
130 135 140
Glu Leu Lys Lys Ala Phe Pro Glu Asn Ile Ser Lys Glu Arg Ser Leu
145 150 155 160
Ile Asn Lys Asn Ile Pro Asn Phe Lys Trp Leu Ala Gly Phe Thr Ser
165 170 175
Gly Glu Gly Cys Phe Phe Val Asn Leu Ile Lys Ser Lys Ser Lys Leu
180 185 190
Gly Val Gln Val Gln Leu Val Phe Ser Ile Thr Gln His Ile Lys Asp
195 200 205
Lys Asn Leu Met Asn Ser Leu Ile Thr Tyr Leu Gly Cys Gly Tyr Ile
210 215 220
Lys Glu Lys Asn Lys Ser Glu Phe Ser Trp Leu Asp Phe Val Val Thr
225 230 235 240
Lys Phe Ser Asp Ile Asn Asp Lys Ile Ile Pro Val Phe Gln Glu Asn
245 250 255
Thr Leu Ile Gly Val Lys Leu Glu Asp Phe Glu Asp Trp Cys Lys Val
260 265 270
Ala Lys Leu Ile Glu Glu Lys Lys His Leu Thr Glu Ser Gly Leu Asp
275 280 285
Glu Ile Lys Lys Ile Lys Leu Asn Met Asn Lys Gly Arg Xaa Xaa
290 295 300
<210>5
<211>303
<212>БЕЛОК
<213>Ophiostoma novo-ulmi subsp.americana (митохондрия)
<220>
<221>MOD_RES
<222>(1)..(8)
<223>Любая аминокислота или отсутствует
<220>
<221>MOD_RES
<222>(302)..(303)
<223>Любая аминокислота или отсутствует
<400>5
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Ser Ile Asn Pro Trp Ile Leu Thr
1 5 10 15
Gly Phe Ala Asp Ala Glu Gly Ser Phe Leu Leu Arg Ile Arg Asn Asn
20 25 30
Asn Lys Ser Ser Val Gly Tyr Ser Thr Glu Leu Gly Phe Gln Ile Thr
35 40 45
Leu His Asn Lys Asp Lys Ser Ile Leu Glu Asn Ile Gln Ser Thr Trp
50 55 60
Lys Val Gly Val Ile Ala Asn Ser Gly Asp Asn Ala Val Ser Leu Lys
65 70 75 80
Val Thr Arg Phe Glu Asp Leu Lys Val Ile Ile Asp His Phe Glu Lys
85 90 95
Tyr Pro Leu Ile Thr Gln Lys Leu Gly Asp Tyr Lys Leu Phe Lys Gln
100 105 110
Ala Phe Ser Val Met Glu Asn Lys Glu His Leu Lys Glu Asn Gly Ile
115 120 125
Lys Glu Leu Val Arg Ile Lys Ala Lys Leu Asn Trp Gly Leu Thr Asp
130 135 140
Glu Leu Lys Lys Ala Phe Pro Glu Asn Ile Ser Lys Glu Arg Ser Leu
145 150 155 160
Ile Asn Lys Asn Ile Pro Asn Phe Lys Trp Leu Ala Gly Phe Thr Ser
165 170 175
Gly Glu Gly Cys Phe Phe Val Asn Leu Ile Lys Ser Lys Ser Lys Leu
180 185 190
Gly Val Gln Val Gln Leu Val Phe Ser Ile Thr Gln His Ile Lys Asp
195 200 205
Lys Asn Leu Met Asn Ser Leu Ile Thr Tyr Leu Gly Cys Gly Tyr Ile
210 215 220
Lys Glu Lys Asn Lys Ser Glu Phe Ser Trp Leu Asp Phe Val Val Thr
225 230 235 240
Lys Phe Ser Asp Ile Asn Asp Lys Ile Ile Pro Val Phe Gln Glu Asn
245 250 255
Thr Leu Ile Gly Val Lys Leu Glu Asp Phe Glu Asp Trp Cys Lys Val
260 265 270
Ala Lys Leu Ile Glu Glu Lys Lys His Leu Thr Glu Ser Gly Leu Asp
275 280 285
Glu Ile Lys Lys Ile Lys Leu Asn Met Asn Lys Gly Arg Xaa Xaa
290 295 300
<210>6
<211>303
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Синтезированный вариант I-OnuI PD-1.ITSM.ex5_RD1_CV3-08
<220>
<221>MOD_RES
<222>(1)..(4)
<223>Любая аминокислота или отсутствует
<220>
<221>MOD_RES
<222>(302)..(303)
<223>Любая аминокислота или отсутствует
<400>6
Xaa Xaa Xaa Xaa Ser Arg Arg Glu Ser Ile Asn Pro Trp Ile Leu Thr
1 5 10 15
Gly Phe Ala Asp Ala Glu Gly Ser Phe Gln Leu Glu Ile Arg Asn Val
20 25 30
Asn Pro Asn Ile Pro Arg Tyr Lys Thr Arg Leu Arg Phe Glu Ile Asp
35 40 45
Leu His Asn Lys Asp Lys Ser Ile Leu Glu Asn Ile Gln Ser Thr Trp
50 55 60
Lys Val Gly Lys Ile Tyr Asn Gln Gly Asp Ser Tyr Val Lys Leu Arg
65 70 75 80
Val Thr Arg Phe Glu Asp Leu Lys Val Ile Ile Asp His Phe Glu Lys
85 90 95
Tyr Pro Leu Ile Thr Gln Lys Leu Gly Asp Tyr Lys Leu Phe Lys Gln
100 105 110
Ala Phe Ser Val Met Glu Asn Lys Glu His Leu Lys Glu Asn Gly Ile
115 120 125
Lys Glu Leu Val Arg Ile Lys Ala Lys Met Asn Trp Gly Leu Asn Asp
130 135 140
Glu Leu Lys Lys Ala Phe Pro Glu Asn Ile Ser Lys Glu Arg Pro Leu
145 150 155 160
Ile Asn Lys Asn Ile Pro Asn Leu Lys Trp Leu Ala Gly Phe Thr Ser
165 170 175
Gly Glu Gly His Phe Gly Val Ile Leu Ala Lys Arg Arg Pro Ala Ser
180 185 190
Pro Val Gln Val Arg Leu Arg Phe Ala Ile Gly Gln His Ile Arg Asp
195 200 205
Lys Asn Leu Met Asn Ser Leu Ile Thr Tyr Leu Gly Cys Gly Arg Ile
210 215 220
Arg Glu Lys Asn Ile Ser Glu Lys Ser Trp Leu Glu Phe Glu Val Thr
225 230 235 240
Lys Phe Ser Asp Ile Asn Asp Lys Ile Ile Pro Val Phe Gln Glu Asn
245 250 255
Thr Leu Ile Gly Val Lys Leu Glu Asp Phe Glu Asp Trp Cys Lys Val
260 265 270
Ala Lys Leu Ile Glu Glu Lys Lys His Leu Thr Glu Ser Gly Leu Asp
275 280 285
Glu Ile Lys Lys Ile Lys Leu Asn Met Asn Lys Gly Arg Xaa Xaa
290 295 300
<210>7
<211>303
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Синтезированный вариант I-OnuI, PD-1.ITSM.ex5_RD2_73
<220>
<221>MOD_RES
<222>(1)..(4)
<223>Любая аминокислота или отсутствует
<220>
<221>MOD_RES
<222>(302)..(303)
<223>Любая аминокислота или отсутствует
<400>7
Xaa Xaa Xaa Xaa Ser Arg Arg Glu Ser Ile Asn Pro Trp Ile Leu Thr
1 5 10 15
Gly Phe Ala Asp Ala Glu Gly Ser Phe Gln Leu Glu Ile Arg Asn Val
20 25 30
Asn Pro Asn Ile Pro Arg Tyr Arg Thr Arg Leu Arg Phe Glu Ile Asp
35 40 45
Leu His Asn Lys Asp Lys Ser Ile Leu Glu Asp Ile Gln Ser Thr Trp
50 55 60
Lys Val Gly Lys Ile Tyr Asn Arg Gly Asp Ser Tyr Val Lys Leu Arg
65 70 75 80
Val Thr Arg Phe Glu Asp Leu Lys Val Ile Ile Asp His Phe Glu Lys
85 90 95
Tyr Pro Leu Ile Thr Gln Lys Leu Gly Asp Tyr Lys Leu Phe Lys Gln
100 105 110
Ala Phe Ser Val Met Glu Asn Lys Glu His Leu Lys Glu Asn Gly Ile
115 120 125
Lys Glu Leu Val Arg Ile Lys Ala Lys Met Asn Trp Gly Leu Asn Asp
130 135 140
Glu Leu Lys Lys Ala Phe Pro Glu Asn Ile Ser Lys Glu Arg Pro Leu
145 150 155 160
Ile Asn Lys Asn Ile Pro Asn Leu Lys Trp Leu Ala Gly Phe Thr Ser
165 170 175
Gly Glu Gly His Phe Gly Val Ile Leu Ala Lys Arg Arg Pro Ala Ser
180 185 190
Pro Val Gln Val Arg Leu Arg Phe Ala Ile Gly Gln His Ile Arg Asp
195 200 205
Lys Asn Leu Met Asn Ser Leu Ile Thr Tyr Leu Gly Cys Gly Arg Thr
210 215 220
Arg Glu Lys Asn Ile Ser Glu Lys Ser Trp Leu Glu Phe Glu Val Thr
225 230 235 240
Lys Phe Ser Asp Ile Asn Asp Lys Ile Ile Pro Val Phe Gln Glu Asn
245 250 255
Thr Leu Ile Gly Val Lys Leu Glu Asp Phe Glu Asp Trp Cys Lys Val
260 265 270
Ala Lys Leu Ile Glu Glu Lys Lys His Leu Thr Glu Ser Gly Leu Asp
275 280 285
Glu Ile Lys Lys Ile Lys Leu Asn Met Asn Lys Gly Arg Xaa Xaa
290 295 300
<210>8
<211>303
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Синтезированный вариант I-OnuI, PD-1.ITSM.ex5_RD3_03
<220>
<221>MOD_RES
<222>(1)..(4)
<223>Любая аминокислота или отсутствует
<220>
<221>MOD_RES
<222>(302)..(303)
<223>Любая аминокислота или отсутствует
<400>8
Xaa Xaa Xaa Xaa Ser Arg Arg Glu Ser Ile Asn Pro Trp Ile Leu Thr
1 5 10 15
Gly Phe Ala Asp Ala Glu Gly Ser Phe Gln Leu Glu Ile Arg Asn Val
20 25 30
Asn Pro Asn Ile Pro Arg Tyr Arg Thr Arg Leu Arg Phe Glu Ile Asp
35 40 45
Leu His Asn Lys Asp Lys Ser Ile Leu Glu Asp Ile Gln Ser Thr Trp
50 55 60
Lys Val Gly Lys Ile Tyr Asn Gln Gly Asp Ser Tyr Val Lys Leu Arg
65 70 75 80
Val Thr Arg Phe Glu Asp Leu Lys Val Ile Ile Asp His Phe Glu Lys
85 90 95
Tyr Pro Leu Ile Thr Gln Lys Leu Gly Asp Tyr Lys Leu Phe Lys Gln
100 105 110
Ala Phe Ser Val Met Glu Asn Lys Glu His Leu Lys Glu Asn Gly Ile
115 120 125
Lys Glu Leu Val Arg Ile Lys Ala Lys Met Asn Trp Gly Leu Asn Asp
130 135 140
Glu Leu Lys Lys Ala Phe Pro Glu Asn Ile Ser Lys Glu Arg Pro Leu
145 150 155 160
Ile Asn Lys Asn Ile Pro Asn Leu Lys Trp Leu Ala Gly Phe Thr Ser
165 170 175
Gly Asp Gly His Phe Gly Val Ile Leu Ala Lys Arg Arg Pro Ala Ser
180 185 190
Pro Val Gln Val Arg Leu Arg Phe Ala Ile Gly Gln His Ile Arg Asp
195 200 205
Lys Asn Leu Met Asn Ser Leu Ile Thr Tyr Leu Gly Cys Gly Arg Thr
210 215 220
Arg Glu Lys Asn Ile Ser Glu Lys Ser Trp Leu Glu Phe Glu Val Thr
225 230 235 240
Lys Phe Ser Asp Ile Asn Asp Lys Ile Ile Pro Val Phe Gln Glu Asn
245 250 255
Thr Leu Ile Gly Val Lys Leu Glu Asp Phe Glu Asp Trp Cys Lys Val
260 265 270
Ala Lys Leu Ile Glu Glu Lys Lys His Leu Thr Glu Ser Gly Leu Asp
275 280 285
Glu Ile Lys Lys Ile Lys Leu Asn Met Asn Lys Gly Arg Xaa Xaa
290 295 300
<210>9
<211>303
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Синтезированный вариант I-OnuI, PD-1.ITSM.ex5_RD4_CV23
<220>
<221>MOD_RES
<222>(1)..(4)
<223>Любая аминокислота или отсутствует
<220>
<221>MOD_RES
<222>(302)..(303)
<223>Любая аминокислота или отсутствует
<400>9
Xaa Xaa Xaa Xaa Ser Arg Arg Glu Ser Ile Asn Pro Trp Ile Leu Thr
1 5 10 15
Gly Phe Ala Asp Ala Glu Gly Ser Phe Gln Leu Glu Ile Arg Asn Val
20 25 30
Asn Pro Asn Ile Pro Arg Tyr Arg Thr Arg Leu Arg Phe Glu Ile Asp
35 40 45
Leu His Asn Lys Asp Lys Ser Ile Leu Glu Asp Ile Gln Ser Thr Trp
50 55 60
Lys Val Gly Lys Ile Tyr Asn Gln Gly Asp Ser Tyr Val Lys Leu Arg
65 70 75 80
Val Thr Arg Phe Glu Asp Leu Lys Val Ile Ile Asp His Phe Glu Lys
85 90 95
Tyr Pro Leu Ile Thr Gln Lys Leu Gly Asp Tyr Lys Leu Phe Lys Gln
100 105 110
Ala Phe Ser Val Met Glu Asn Lys Glu His Leu Lys Glu Asn Gly Ile
115 120 125
Lys Glu Leu Val Arg Ile Lys Ala Lys Met Asn Trp Gly Leu Asn Asp
130 135 140
Glu Leu Lys Lys Ala Phe Pro Glu Asn Ile Ser Lys Glu Arg Pro Leu
145 150 155 160
Ile Asn Lys Asn Ile Pro Asn Leu Lys Trp Leu Ala Gly Phe Thr Ser
165 170 175
Gly Asp Gly His Phe Gly Val Ile Leu Ala Lys Arg Arg Pro Ala Ser
180 185 190
Pro Val Gln Val Arg Leu Arg Phe Ala Ile Gly Gln His Ile Arg Asp
195 200 205
Lys Asn Leu Met Asn Ser Leu Ile Thr Tyr Leu Gly Cys Gly Arg Ile
210 215 220
Arg Glu Lys Asn Lys Ser Glu Lys Ser Trp Leu Glu Phe Glu Val Thr
225 230 235 240
Lys Phe Ser Asp Ile Asn Asp Lys Ile Ile Pro Val Phe Gln Glu Asn
245 250 255
Thr Leu Ile Gly Val Lys Leu Glu Asp Phe Glu Asp Trp Cys Lys Val
260 265 270
Ala Lys Leu Ile Glu Glu Lys Lys His Leu Thr Glu Ser Gly Leu Asp
275 280 285
Glu Ile Lys Lys Ile Lys Leu Asn Met Asn Lys Gly Arg Xaa Xaa
290 295 300
<210>10
<211>303
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Синтезированный вариант I-OnuI, PD-1.ITSM.ex5_RD5_CV23MK
<220>
<221>MOD_RES
<222>(1)..(4)
<223>Любая аминокислота или отсутствует
<220>
<221>MOD_RES
<222>(302)..(303)
<223>Любая аминокислота или отсутствует
<400>10
Xaa Xaa Xaa Xaa Ser Arg Arg Glu Ser Ile Asn Pro Trp Ile Leu Thr
1 5 10 15
Gly Phe Ala Asp Ala Glu Gly Ser Phe Gln Leu Glu Ile Arg Asn Val
20 25 30
Asn Pro Asn Ile Pro Arg Tyr Arg Thr Arg Leu Arg Phe Glu Ile Asp
35 40 45
Leu His Asn Lys Asp Lys Ser Ile Leu Glu Asp Ile Gln Ser Thr Trp
50 55 60
Lys Val Gly Lys Ile Tyr Asn Gln Gly Asp Ser Tyr Val Lys Leu Arg
65 70 75 80
Val Thr Arg Phe Glu Asp Leu Lys Val Ile Ile Asp His Phe Glu Lys
85 90 95
Tyr Pro Leu Ile Thr Gln Lys Leu Gly Asp Tyr Lys Leu Phe Lys Gln
100 105 110
Ala Phe Ser Val Met Glu Asn Lys Glu His Leu Lys Glu Asn Gly Ile
115 120 125
Lys Glu Leu Val Arg Ile Lys Ala Lys Met Asn Trp Gly Leu Asn Asp
130 135 140
Glu Leu Lys Lys Ala Phe Pro Glu Asn Ile Ser Lys Glu Arg Pro Leu
145 150 155 160
Ile Asn Lys Asn Ile Pro Asn Leu Lys Trp Leu Ala Gly Phe Thr Ser
165 170 175
Gly Asp Gly His Phe Gly Val Ile Leu Ala Lys Arg Arg Pro Ala Ser
180 185 190
Pro Val Gln Val Arg Leu Arg Phe Met Ile Gly Gln His Ile Lys Asp
195 200 205
Lys Asn Leu Met Asn Ser Leu Ile Thr Tyr Leu Gly Cys Gly Arg Ile
210 215 220
Arg Glu Lys Asn Lys Ser Glu Lys Ser Trp Leu Glu Phe Glu Val Thr
225 230 235 240
Lys Phe Ser Asp Ile Asn Asp Lys Ile Ile Pro Val Phe Gln Glu Asn
245 250 255
Thr Leu Ile Gly Val Lys Leu Glu Asp Phe Glu Asp Trp Cys Lys Val
260 265 270
Ala Lys Leu Ile Glu Glu Lys Lys His Leu Thr Glu Ser Gly Leu Asp
275 280 285
Glu Ile Lys Lys Ile Lys Leu Asn Met Asn Lys Gly Arg Xaa Xaa
290 295 300
<210>11
<211>303
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Синтезированный вариант I-OnuI, PD-1.ile3.exon1_RD1_B1
<220>
<221>MOD_RES
<222>(1)..(4)
<223>Любая аминокислота или отсутствует
<220>
<221>MOD_RES
<222>(302)..(303)
<223>Любая аминокислота или отсутствует
<400>11
Xaa Xaa Xaa Xaa Ser Arg Arg Glu Ser Ile Asn Pro Trp Ile Leu Thr
1 5 10 15
Gly Phe Ala Asp Ala Glu Gly Ser Phe Gly Leu Ser Ile Leu Asn Arg
20 25 30
Asn Arg Gly Thr Ala Arg Tyr His Thr Arg Leu Ser Phe Ala Ile Val
35 40 45
Leu His Asn Lys Asp Lys Ser Ile Leu Glu Asn Ile Gln Ser Thr Trp
50 55 60
Lys Val Gly Ile Ile Thr Asn Asp Gly Asp Arg Tyr Val Arg Leu Arg
65 70 75 80
Val Thr Arg Phe Glu Asp Leu Lys Val Ile Ile Asp His Phe Glu Lys
85 90 95
Tyr Pro Leu Val Thr Gln Lys Leu Gly Asp Tyr Lys Leu Phe Lys Gln
100 105 110
Ala Phe Ser Val Met Glu Asn Lys Glu His Leu Lys Glu Asn Gly Ile
115 120 125
Lys Glu Leu Val Arg Ile Lys Ala Lys Met Asn Trp Gly Leu Asn Asp
130 135 140
Glu Leu Lys Lys Ala Phe Pro Glu Asn Ile Ser Lys Glu Arg Pro Leu
145 150 155 160
Ile Asn Lys Asn Ile Pro Asn Phe Lys Trp Leu Ala Gly Phe Thr Ser
165 170 175
Gly Asp Gly Ser Phe Phe Val Arg Leu Arg Lys Ser Asn Val Asn Ala
180 185 190
Arg Val Arg Val Gln Leu Val Phe Glu Ile Ser Gln His Ile Arg Asp
195 200 205
Lys Asn Leu Met Asn Ser Leu Ile Thr Tyr Leu Gly Cys Gly His Ile
210 215 220
Tyr Glu Gly Asn Lys Ser Glu Arg Ser Trp Leu Gln Phe Arg Val Glu
225 230 235 240
Lys Phe Ser Asp Ile Asn Asp Lys Ile Ile Pro Val Phe Gln Glu Asn
245 250 255
Thr Leu Ile Gly Val Lys Leu Glu Asp Phe Glu Asp Trp Cys Lys Val
260 265 270
Ala Lys Leu Ile Glu Glu Lys Lys His Leu Thr Glu Ser Gly Leu Asp
275 280 285
Glu Ile Lys Lys Ile Lys Leu Asn Met Asn Lys Gly Arg Xaa Xaa
290 295 300
<210>12
<211>303
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Синтезированный вариант I-OnuI, PD-1.ile3.exon1_RD2_B1H8
<220>
<221>MOD_RES
<222>(1)..(4)
<223>Любая аминокислота или отсутствует
<220>
<221>MOD_RES
<222>(302)..(303)
<223>Любая аминокислота или отсутствует
<400>12
Xaa Xaa Xaa Xaa Ser Arg Arg Glu Ser Ile Asn Pro Trp Ile Leu Thr
1 5 10 15
Gly Phe Ala Asp Ala Glu Gly Ser Phe Gly Leu Ser Ile Leu Asn Arg
20 25 30
Asn Arg Gly Thr Ala Arg Tyr His Thr Arg Leu Ser Phe Thr Ile Val
35 40 45
Leu His Asn Lys Asp Lys Ser Ile Leu Glu Asn Ile Gln Ser Thr Trp
50 55 60
Lys Val Gly Ile Ile Thr Asn Asp Gly Asp Arg Tyr Val Arg Leu Cys
65 70 75 80
Val Thr Arg Phe Glu Asp Leu Lys Val Ile Ile Asp His Phe Glu Lys
85 90 95
Tyr Pro Leu Val Thr Gln Lys Leu Gly Asp Tyr Lys Leu Phe Lys Gln
100 105 110
Ala Phe Ser Val Met Glu Asn Lys Glu His Leu Lys Glu Asn Gly Ile
115 120 125
Lys Glu Leu Ala Arg Ile Lys Ala Lys Met Asn Trp Gly Leu Asn Asp
130 135 140
Glu Leu Lys Lys Ala Phe Pro Glu Asn Ile Gly Lys Glu Arg Pro Leu
145 150 155 160
Ile Asn Lys Asn Ile Pro Asn Phe Lys Trp Leu Ala Gly Phe Thr Ser
165 170 175
Gly Asp Gly Ser Phe Phe Val Arg Leu Arg Lys Ser Asn Val Asn Ala
180 185 190
Arg Val Arg Val Gln Leu Val Phe Glu Ile Ser Gln His Ile Arg Asp
195 200 205
Lys Asn Leu Met Asn Ser Leu Ile Thr Tyr Leu Gly Cys Gly His Ile
210 215 220
Tyr Glu Gly Asn Lys Ser Glu Arg Ser Trp Leu Gln Phe Arg Val Glu
225 230 235 240
Lys Phe Ser Asp Ile Asn Asp Lys Ile Ile Pro Val Phe Gln Glu Asn
245 250 255
Thr Leu Ile Gly Val Lys Leu Glu Asp Phe Glu Asp Trp Cys Lys Val
260 265 270
Ala Lys Leu Ile Glu Glu Lys Lys His Leu Thr Glu Ser Gly Leu Asp
275 280 285
Glu Ile Lys Lys Ile Lys Leu Asn Met Asn Lys Gly Arg Xaa Xaa
290 295 300
<210>13
<211>303
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Синтезированный вариант I-OnuI, PD-1.IgV.exon2_RD1_G5
<220>
<221>MOD_RES
<222>(1)..(4)
<223>Любая аминокислота или отсутствует
<220>
<221>MOD_RES
<222>(302)..(303)
<223>Любая аминокислота или отсутствует
<400>13
Xaa Xaa Xaa Xaa Ser Arg Arg Glu Ser Ile Asn Pro Trp Ile Leu Thr
1 5 10 15
Gly Phe Ala Asp Ala Glu Gly Ser Phe Gln Leu Tyr Ile Ser Asn Val
20 25 30
Asn Asn Asn Arg Ser Arg Tyr Arg Ala Arg Leu Arg Phe Ala Ile Glu
35 40 45
Leu His Asn Lys Asp Lys Ser Ile Leu Glu Asn Ile Gln Ser Thr Trp
50 55 60
Lys Val Gly Val Ile Asn Asn Ile Gly Asp Thr Ser Val Arg Leu Ser
65 70 75 80
Val Gly Arg Phe Glu Asp Leu Lys Val Ile Ile Asp His Phe Glu Lys
85 90 95
Tyr Pro Leu Ile Thr Gln Lys Leu Gly Asp Tyr Lys Leu Phe Lys Gln
100 105 110
Ala Phe Ser Val Met Glu Asn Lys Glu His Leu Lys Glu Asn Gly Ile
115 120 125
Lys Glu Leu Val Arg Ile Lys Ala Lys Met Asn Trp Gly Leu Asn Asp
130 135 140
Glu Leu Lys Lys Ala Phe Pro Glu Asn Ile Ser Lys Glu Arg Pro Leu
145 150 155 160
Ile Asn Lys Asn Ile Pro Asn Leu Lys Trp Leu Ala Gly Phe Thr Ser
165 170 175
Gly Asp Gly Asn Phe Tyr Val His Leu Lys Lys Ser Gly Arg Thr Thr
180 185 190
Arg Val Tyr Val Gln Leu Arg Phe Ser Ile Ala Gln His Ile Arg Asp
195 200 205
Lys Asn Leu Met Asn Ser Leu Ile Thr Tyr Leu Gly Cys Gly Tyr Ile
210 215 220
Asn Glu Trp Asn Ala Ser Glu Arg Ser Ala Leu Glu Phe Arg Val Thr
225 230 235 240
Lys Phe Ser Asp Ile Asn Asp Lys Ile Ile Pro Val Phe Gln Glu Asn
245 250 255
Thr Leu Ile Gly Val Lys Leu Glu Asp Phe Glu Asp Trp Cys Lys Val
260 265 270
Ala Lys Leu Ile Glu Glu Lys Lys His Leu Thr Glu Ser Gly Leu Asp
275 280 285
Glu Ile Lys Lys Ile Lys Leu Asn Met Asn Lys Gly Arg Xaa Xaa
290 295 300
<210>14
<211>303
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Синтезированный вариант I-OnuI, PD-1.IgV.exon2_RD1_PS3
<220>
<221>MOD_RES
<222>(1)..(4)
<223>Любая аминокислота или отсутствует
<220>
<221>MOD_RES
<222>(302)..(303)
<223>Любая аминокислота или отсутствует
<400>14
Xaa Xaa Xaa Xaa Ser Arg Arg Glu Ser Ile Asn Pro Trp Ile Leu Thr
1 5 10 15
Gly Phe Ala Asp Ala Glu Gly Cys Phe Leu Leu His Ile Arg Asn Leu
20 25 30
Asn Arg Thr Ser Thr Lys Tyr Arg Thr Arg Leu Ser Phe Glu Ile Glu
35 40 45
Leu His Asn Lys Asp Lys Ser Ile Leu Glu Asn Ile Gln Ser Thr Trp
50 55 60
Lys Val Gly Ile Ile Asn Asn Ile Gly Asn Arg Arg Val Arg Leu Ser
65 70 75 80
Val Arg Arg Phe Glu Asp Leu Lys Val Ile Ile Asp His Phe Glu Lys
85 90 95
Tyr Pro Leu Ile Thr Gln Lys Leu Gly Asp Tyr Lys Leu Phe Lys Gln
100 105 110
Ala Phe Ser Leu Met Glu Asn Lys Glu His Leu Lys Glu Asn Gly Ile
115 120 125
Lys Glu Leu Val Arg Ile Lys Ala Lys Met Asn Trp Gly Leu Asn Asp
130 135 140
Glu Leu Lys Lys Ala Phe Pro Glu Asn Ile Ser Lys Glu Arg Pro Leu
145 150 155 160
Ile Asn Lys Asn Ile Pro Asn Leu Lys Trp Leu Ala Gly Phe Thr Ser
165 170 175
Gly Asp Gly Asn Phe Tyr Val His Leu Lys Lys Ser Gly Arg Thr Thr
180 185 190
Arg Val Tyr Val Gln Leu Arg Phe Ser Ile Ser Gln His Ile Arg Asp
195 200 205
Lys Asn Leu Met Asn Ser Leu Ile Thr Tyr Leu Gly Cys Gly Tyr Ile
210 215 220
Thr Glu Ser Asn Pro Ser Glu Arg Ser Asp Leu Glu Phe Arg Val Thr
225 230 235 240
Lys Phe Ser Asp Ile Asn Asp Lys Ile Ile Pro Val Phe Gln Glu Asn
245 250 255
Thr Leu Ile Gly Val Lys Leu Glu Asp Phe Glu Asp Trp Cys Lys Val
260 265 270
Ala Lys Leu Ile Glu Glu Lys Lys His Leu Thr Glu Ser Gly Leu Asp
275 280 285
Glu Ile Lys Lys Ile Lys Leu Asn Met Asn Lys Gly Arg Xaa Xaa
290 295 300
<210>15
<211>870
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Синтезированный megaTAL PD-1.ITSM.ex5_RD1_CV3-08
<400>15
Met Gly Ser Ala Pro Pro Lys Lys Lys Arg Lys Val Val Asp Leu Arg
1 5 10 15
Thr Leu Gly Tyr Ser Gln Gln Gln Gln Glu Lys Ile Lys Pro Lys Val
20 25 30
Arg Ser Thr Val Ala Gln His His Glu Ala Leu Val Gly His Gly Phe
35 40 45
Thr His Ala His Ile Val Ala Leu Ser Gln His Pro Ala Ala Leu Gly
50 55 60
Thr Val Ala Val Thr Tyr Gln His Ile Ile Thr Ala Leu Pro Glu Ala
65 70 75 80
Thr His Glu Asp Ile Val Gly Val Gly Lys Gln Trp Ser Gly Ala Arg
85 90 95
Ala Leu Glu Ala Leu Leu Thr Asp Ala Gly Glu Leu Arg Gly Pro Pro
100 105 110
Leu Gln Leu Asp Thr Gly Gln Leu Val Lys Ile Ala Lys Arg Gly Gly
115 120 125
Val Thr Ala Met Glu Ala Val His Ala Ser Arg Asn Ala Leu Thr Gly
130 135 140
Ala Pro Leu Asn Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn
145 150 155 160
Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
165 170 175
Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala
180 185 190
Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
195 200 205
Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala
210 215 220
Ile Ala Ser Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu
225 230 235 240
Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val
245 250 255
Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln
260 265 270
Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln
275 280 285
Val Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr
290 295 300
Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro
305 310 315 320
Asp Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu
325 330 335
Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu
340 345 350
Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn Ile Gly Gly Lys Gln
355 360 365
Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His
370 375 380
Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly
385 390 395 400
Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln
405 410 415
Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn Asn
420 425 430
Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu
435 440 445
Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser
450 455 460
Asn Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro
465 470 475 480
Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile
485 490 495
Ala Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu Ser Ile Val Ala Gln
500 505 510
Leu Ser Arg Pro Asp Pro Ala Leu Ala Ala Leu Thr Asn Asp His Leu
515 520 525
Val Ala Leu Ala Cys Leu Gly Gly Arg Pro Ala Met Asp Ala Val Lys
530 535 540
Lys Gly Leu Pro His Ala Pro Glu Leu Ile Arg Arg Val Asn Arg Arg
545 550 555 560
Ile Gly Glu Arg Thr Ser His Arg Val Ala Ile Ser Arg Ser Arg Arg
565 570 575
Glu Ser Ile Asn Pro Trp Ile Leu Thr Gly Phe Ala Asp Ala Glu Gly
580 585 590
Ser Phe Gln Leu Glu Ile Arg Asn Val Asn Pro Asn Ile Pro Arg Tyr
595 600 605
Lys Thr Arg Leu Arg Phe Glu Ile Asp Leu His Asn Lys Asp Lys Ser
610 615 620
Ile Leu Glu Asn Ile Gln Ser Thr Trp Lys Val Gly Lys Ile Tyr Asn
625 630 635 640
Gln Gly Asp Ser Tyr Val Lys Leu Arg Val Thr Arg Phe Glu Asp Leu
645 650 655
Lys Val Ile Ile Asp His Phe Glu Lys Tyr Pro Leu Ile Thr Gln Lys
660 665 670
Leu Gly Asp Tyr Lys Leu Phe Lys Gln Ala Phe Ser Val Met Glu Asn
675 680 685
Lys Glu His Leu Lys Glu Asn Gly Ile Lys Glu Leu Val Arg Ile Lys
690 695 700
Ala Lys Met Asn Trp Gly Leu Asn Asp Glu Leu Lys Lys Ala Phe Pro
705 710 715 720
Glu Asn Ile Ser Lys Glu Arg Pro Leu Ile Asn Lys Asn Ile Pro Asn
725 730 735
Leu Lys Trp Leu Ala Gly Phe Thr Ser Gly Glu Gly His Phe Gly Val
740 745 750
Ile Leu Ala Lys Arg Arg Pro Ala Ser Pro Val Gln Val Arg Leu Arg
755 760 765
Phe Ala Ile Gly Gln His Ile Arg Asp Lys Asn Leu Met Asn Ser Leu
770 775 780
Ile Thr Tyr Leu Gly Cys Gly Arg Ile Arg Glu Lys Asn Ile Ser Glu
785 790 795 800
Lys Ser Trp Leu Glu Phe Glu Val Thr Lys Phe Ser Asp Ile Asn Asp
805 810 815
Lys Ile Ile Pro Val Phe Gln Glu Asn Thr Leu Ile Gly Val Lys Leu
820 825 830
Glu Asp Phe Glu Asp Trp Cys Lys Val Ala Lys Leu Ile Glu Glu Lys
835 840 845
Lys His Leu Thr Glu Ser Gly Leu Asp Glu Ile Lys Lys Ile Lys Leu
850 855 860
Asn Met Asn Lys Gly Arg
865 870
<210>16
<211>870
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Синтезированный megaTAL PD-1.ITSM.ex5_RD2_73
<400>16
Met Gly Ser Ala Pro Pro Lys Lys Lys Arg Lys Val Val Asp Leu Arg
1 5 10 15
Thr Leu Gly Tyr Ser Gln Gln Gln Gln Glu Lys Ile Lys Pro Lys Val
20 25 30
Arg Ser Thr Val Ala Gln His His Glu Ala Leu Val Gly His Gly Phe
35 40 45
Thr His Ala His Ile Val Ala Leu Ser Gln His Pro Ala Ala Leu Gly
50 55 60
Thr Val Ala Val Thr Tyr Gln His Ile Ile Thr Ala Leu Pro Glu Ala
65 70 75 80
Thr His Glu Asp Ile Val Gly Val Gly Lys Gln Trp Ser Gly Ala Arg
85 90 95
Ala Leu Glu Ala Leu Leu Thr Asp Ala Gly Glu Leu Arg Gly Pro Pro
100 105 110
Leu Gln Leu Asp Thr Gly Gln Leu Val Lys Ile Ala Lys Arg Gly Gly
115 120 125
Val Thr Ala Met Glu Ala Val His Ala Ser Arg Asn Ala Leu Thr Gly
130 135 140
Ala Pro Leu Asn Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn
145 150 155 160
Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
165 170 175
Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala
180 185 190
Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
195 200 205
Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala
210 215 220
Ile Ala Ser Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu
225 230 235 240
Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val
245 250 255
Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln
260 265 270
Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln
275 280 285
Val Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr
290 295 300
Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro
305 310 315 320
Asp Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu
325 330 335
Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu
340 345 350
Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn Ile Gly Gly Lys Gln
355 360 365
Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His
370 375 380
Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly
385 390 395 400
Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln
405 410 415
Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn Asn
420 425 430
Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu
435 440 445
Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser
450 455 460
Asn Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro
465 470 475 480
Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile
485 490 495
Ala Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu Ser Ile Val Ala Gln
500 505 510
Leu Ser Arg Pro Asp Pro Ala Leu Ala Ala Leu Thr Asn Asp His Leu
515 520 525
Val Ala Leu Ala Cys Leu Gly Gly Arg Pro Ala Met Asp Ala Val Lys
530 535 540
Lys Gly Leu Pro His Ala Pro Glu Leu Ile Arg Arg Val Asn Arg Arg
545 550 555 560
Ile Gly Glu Arg Thr Ser His Arg Val Ala Ile Ser Arg Ser Arg Arg
565 570 575
Glu Ser Ile Asn Pro Trp Ile Leu Thr Gly Phe Ala Asp Ala Glu Gly
580 585 590
Ser Phe Gln Leu Glu Ile Arg Asn Val Asn Pro Asn Ile Pro Arg Tyr
595 600 605
Arg Thr Arg Leu Arg Phe Glu Ile Asp Leu His Asn Lys Asp Lys Ser
610 615 620
Ile Leu Glu Asp Ile Gln Ser Thr Trp Lys Val Gly Lys Ile Tyr Asn
625 630 635 640
Arg Gly Asp Ser Tyr Val Lys Leu Arg Val Thr Arg Phe Glu Asp Leu
645 650 655
Lys Val Ile Ile Asp His Phe Glu Lys Tyr Pro Leu Ile Thr Gln Lys
660 665 670
Leu Gly Asp Tyr Lys Leu Phe Lys Gln Ala Phe Ser Val Met Glu Asn
675 680 685
Lys Glu His Leu Lys Glu Asn Gly Ile Lys Glu Leu Val Arg Ile Lys
690 695 700
Ala Lys Met Asn Trp Gly Leu Asn Asp Glu Leu Lys Lys Ala Phe Pro
705 710 715 720
Glu Asn Ile Ser Lys Glu Arg Pro Leu Ile Asn Lys Asn Ile Pro Asn
725 730 735
Leu Lys Trp Leu Ala Gly Phe Thr Ser Gly Glu Gly His Phe Gly Val
740 745 750
Ile Leu Ala Lys Arg Arg Pro Ala Ser Pro Val Gln Val Arg Leu Arg
755 760 765
Phe Ala Ile Gly Gln His Ile Arg Asp Lys Asn Leu Met Asn Ser Leu
770 775 780
Ile Thr Tyr Leu Gly Cys Gly Arg Thr Arg Glu Lys Asn Ile Ser Glu
785 790 795 800
Lys Ser Trp Leu Glu Phe Glu Val Thr Lys Phe Ser Asp Ile Asn Asp
805 810 815
Lys Ile Ile Pro Val Phe Gln Glu Asn Thr Leu Ile Gly Val Lys Leu
820 825 830
Glu Asp Phe Glu Asp Trp Cys Lys Val Ala Lys Leu Ile Glu Glu Lys
835 840 845
Lys His Leu Thr Glu Ser Gly Leu Asp Glu Ile Lys Lys Ile Lys Leu
850 855 860
Asn Met Asn Lys Gly Arg
865 870
<210>17
<211>870
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Синтезированный megaTAL PD-1.ITSM.ex5_RD3_03
<400>17
Met Gly Ser Ala Pro Pro Lys Lys Lys Arg Lys Val Val Asp Leu Arg
1 5 10 15
Thr Leu Gly Tyr Ser Gln Gln Gln Gln Glu Lys Ile Lys Pro Lys Val
20 25 30
Arg Ser Thr Val Ala Gln His His Glu Ala Leu Val Gly His Gly Phe
35 40 45
Thr His Ala His Ile Val Ala Leu Ser Gln His Pro Ala Ala Leu Gly
50 55 60
Thr Val Ala Val Thr Tyr Gln His Ile Ile Thr Ala Leu Pro Glu Ala
65 70 75 80
Thr His Glu Asp Ile Val Gly Val Gly Lys Gln Trp Ser Gly Ala Arg
85 90 95
Ala Leu Glu Ala Leu Leu Thr Asp Ala Gly Glu Leu Arg Gly Pro Pro
100 105 110
Leu Gln Leu Asp Thr Gly Gln Leu Val Lys Ile Ala Lys Arg Gly Gly
115 120 125
Val Thr Ala Met Glu Ala Val His Ala Ser Arg Asn Ala Leu Thr Gly
130 135 140
Ala Pro Leu Asn Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn
145 150 155 160
Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
165 170 175
Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala
180 185 190
Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
195 200 205
Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala
210 215 220
Ile Ala Ser Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu
225 230 235 240
Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val
245 250 255
Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln
260 265 270
Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln
275 280 285
Val Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr
290 295 300
Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro
305 310 315 320
Asp Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu
325 330 335
Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu
340 345 350
Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn Ile Gly Gly Lys Gln
355 360 365
Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His
370 375 380
Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly
385 390 395 400
Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln
405 410 415
Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn Asn
420 425 430
Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu
435 440 445
Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser
450 455 460
Asn Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro
465 470 475 480
Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile
485 490 495
Ala Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu Ser Ile Val Ala Gln
500 505 510
Leu Ser Arg Pro Asp Pro Ala Leu Ala Ala Leu Thr Asn Asp His Leu
515 520 525
Val Ala Leu Ala Cys Leu Gly Gly Arg Pro Ala Met Asp Ala Val Lys
530 535 540
Lys Gly Leu Pro His Ala Pro Glu Leu Ile Arg Arg Val Asn Arg Arg
545 550 555 560
Ile Gly Glu Arg Thr Ser His Arg Val Ala Ile Ser Arg Ser Arg Arg
565 570 575
Glu Ser Ile Asn Pro Trp Ile Leu Thr Gly Phe Ala Asp Ala Glu Gly
580 585 590
Ser Phe Gln Leu Glu Ile Arg Asn Val Asn Pro Asn Ile Pro Arg Tyr
595 600 605
Arg Thr Arg Leu Arg Phe Glu Ile Asp Leu His Asn Lys Asp Lys Ser
610 615 620
Ile Leu Glu Asp Ile Gln Ser Thr Trp Lys Val Gly Lys Ile Tyr Asn
625 630 635 640
Gln Gly Asp Ser Tyr Val Lys Leu Arg Val Thr Arg Phe Glu Asp Leu
645 650 655
Lys Val Ile Ile Asp His Phe Glu Lys Tyr Pro Leu Ile Thr Gln Lys
660 665 670
Leu Gly Asp Tyr Lys Leu Phe Lys Gln Ala Phe Ser Val Met Glu Asn
675 680 685
Lys Glu His Leu Lys Glu Asn Gly Ile Lys Glu Leu Val Arg Ile Lys
690 695 700
Ala Lys Met Asn Trp Gly Leu Asn Asp Glu Leu Lys Lys Ala Phe Pro
705 710 715 720
Glu Asn Ile Ser Lys Glu Arg Pro Leu Ile Asn Lys Asn Ile Pro Asn
725 730 735
Leu Lys Trp Leu Ala Gly Phe Thr Ser Gly Asp Gly His Phe Gly Val
740 745 750
Ile Leu Ala Lys Arg Arg Pro Ala Ser Pro Val Gln Val Arg Leu Arg
755 760 765
Phe Ala Ile Gly Gln His Ile Arg Asp Lys Asn Leu Met Asn Ser Leu
770 775 780
Ile Thr Tyr Leu Gly Cys Gly Arg Thr Arg Glu Lys Asn Ile Ser Glu
785 790 795 800
Lys Ser Trp Leu Glu Phe Glu Val Thr Lys Phe Ser Asp Ile Asn Asp
805 810 815
Lys Ile Ile Pro Val Phe Gln Glu Asn Thr Leu Ile Gly Val Lys Leu
820 825 830
Glu Asp Phe Glu Asp Trp Cys Lys Val Ala Lys Leu Ile Glu Glu Lys
835 840 845
Lys His Leu Thr Glu Ser Gly Leu Asp Glu Ile Lys Lys Ile Lys Leu
850 855 860
Asn Met Asn Lys Gly Arg
865 870
<210>18
<211>870
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Синтезированный megaTAL PD-1.ITSM.ex5_RD4_CV23
<400>18
Met Gly Ser Ala Pro Pro Lys Lys Lys Arg Lys Val Val Asp Leu Arg
1 5 10 15
Thr Leu Gly Tyr Ser Gln Gln Gln Gln Glu Lys Ile Lys Pro Lys Val
20 25 30
Arg Ser Thr Val Ala Gln His His Glu Ala Leu Val Gly His Gly Phe
35 40 45
Thr His Ala His Ile Val Ala Leu Ser Gln His Pro Ala Ala Leu Gly
50 55 60
Thr Val Ala Val Thr Tyr Gln His Ile Ile Thr Ala Leu Pro Glu Ala
65 70 75 80
Thr His Glu Asp Ile Val Gly Val Gly Lys Gln Trp Ser Gly Ala Arg
85 90 95
Ala Leu Glu Ala Leu Leu Thr Asp Ala Gly Glu Leu Arg Gly Pro Pro
100 105 110
Leu Gln Leu Asp Thr Gly Gln Leu Val Lys Ile Ala Lys Arg Gly Gly
115 120 125
Val Thr Ala Met Glu Ala Val His Ala Ser Arg Asn Ala Leu Thr Gly
130 135 140
Ala Pro Leu Asn Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn
145 150 155 160
Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
165 170 175
Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala
180 185 190
Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
195 200 205
Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala
210 215 220
Ile Ala Ser Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu
225 230 235 240
Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val
245 250 255
Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln
260 265 270
Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln
275 280 285
Val Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr
290 295 300
Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro
305 310 315 320
Asp Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu
325 330 335
Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu
340 345 350
Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn Ile Gly Gly Lys Gln
355 360 365
Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His
370 375 380
Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly
385 390 395 400
Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln
405 410 415
Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn Asn
420 425 430
Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu
435 440 445
Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser
450 455 460
Asn Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro
465 470 475 480
Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile
485 490 495
Ala Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu Ser Ile Val Ala Gln
500 505 510
Leu Ser Arg Pro Asp Pro Ala Leu Ala Ala Leu Thr Asn Asp His Leu
515 520 525
Val Ala Leu Ala Cys Leu Gly Gly Arg Pro Ala Met Asp Ala Val Lys
530 535 540
Lys Gly Leu Pro His Ala Pro Glu Leu Ile Arg Arg Val Asn Arg Arg
545 550 555 560
Ile Gly Glu Arg Thr Ser His Arg Val Ala Ile Ser Arg Ser Arg Arg
565 570 575
Glu Ser Ile Asn Pro Trp Ile Leu Thr Gly Phe Ala Asp Ala Glu Gly
580 585 590
Ser Phe Gln Leu Glu Ile Arg Asn Val Asn Pro Asn Ile Pro Arg Tyr
595 600 605
Arg Thr Arg Leu Arg Phe Glu Ile Asp Leu His Asn Lys Asp Lys Ser
610 615 620
Ile Leu Glu Asp Ile Gln Ser Thr Trp Lys Val Gly Lys Ile Tyr Asn
625 630 635 640
Gln Gly Asp Ser Tyr Val Lys Leu Arg Val Thr Arg Phe Glu Asp Leu
645 650 655
Lys Val Ile Ile Asp His Phe Glu Lys Tyr Pro Leu Ile Thr Gln Lys
660 665 670
Leu Gly Asp Tyr Lys Leu Phe Lys Gln Ala Phe Ser Val Met Glu Asn
675 680 685
Lys Glu His Leu Lys Glu Asn Gly Ile Lys Glu Leu Val Arg Ile Lys
690 695 700
Ala Lys Met Asn Trp Gly Leu Asn Asp Glu Leu Lys Lys Ala Phe Pro
705 710 715 720
Glu Asn Ile Ser Lys Glu Arg Pro Leu Ile Asn Lys Asn Ile Pro Asn
725 730 735
Leu Lys Trp Leu Ala Gly Phe Thr Ser Gly Asp Gly His Phe Gly Val
740 745 750
Ile Leu Ala Lys Arg Arg Pro Ala Ser Pro Val Gln Val Arg Leu Arg
755 760 765
Phe Ala Ile Gly Gln His Ile Arg Asp Lys Asn Leu Met Asn Ser Leu
770 775 780
Ile Thr Tyr Leu Gly Cys Gly Arg Ile Arg Glu Lys Asn Lys Ser Glu
785 790 795 800
Lys Ser Trp Leu Glu Phe Glu Val Thr Lys Phe Ser Asp Ile Asn Asp
805 810 815
Lys Ile Ile Pro Val Phe Gln Glu Asn Thr Leu Ile Gly Val Lys Leu
820 825 830
Glu Asp Phe Glu Asp Trp Cys Lys Val Ala Lys Leu Ile Glu Glu Lys
835 840 845
Lys His Leu Thr Glu Ser Gly Leu Asp Glu Ile Lys Lys Ile Lys Leu
850 855 860
Asn Met Asn Lys Gly Arg
865 870
<210>19
<211>870
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Синтезированный megaTAL PD-1.ITSM.ex5_RD5_CV23MK
<400>19
Met Gly Ser Ala Pro Pro Lys Lys Lys Arg Lys Val Val Asp Leu Arg
1 5 10 15
Thr Leu Gly Tyr Ser Gln Gln Gln Gln Glu Lys Ile Lys Pro Lys Val
20 25 30
Arg Ser Thr Val Ala Gln His His Glu Ala Leu Val Gly His Gly Phe
35 40 45
Thr His Ala His Ile Val Ala Leu Ser Gln His Pro Ala Ala Leu Gly
50 55 60
Thr Val Ala Val Thr Tyr Gln His Ile Ile Thr Ala Leu Pro Glu Ala
65 70 75 80
Thr His Glu Asp Ile Val Gly Val Gly Lys Gln Trp Ser Gly Ala Arg
85 90 95
Ala Leu Glu Ala Leu Leu Thr Asp Ala Gly Glu Leu Arg Gly Pro Pro
100 105 110
Leu Gln Leu Asp Thr Gly Gln Leu Val Lys Ile Ala Lys Arg Gly Gly
115 120 125
Val Thr Ala Met Glu Ala Val His Ala Ser Arg Asn Ala Leu Thr Gly
130 135 140
Ala Pro Leu Asn Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn
145 150 155 160
Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
165 170 175
Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala
180 185 190
Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
195 200 205
Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala
210 215 220
Ile Ala Ser Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu
225 230 235 240
Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val
245 250 255
Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln
260 265 270
Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln
275 280 285
Val Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr
290 295 300
Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro
305 310 315 320
Asp Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu
325 330 335
Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu
340 345 350
Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn Ile Gly Gly Lys Gln
355 360 365
Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His
370 375 380
Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly
385 390 395 400
Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln
405 410 415
Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn Asn
420 425 430
Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu
435 440 445
Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser
450 455 460
Asn Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro
465 470 475 480
Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile
485 490 495
Ala Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu Ser Ile Val Ala Gln
500 505 510
Leu Ser Arg Pro Asp Pro Ala Leu Ala Ala Leu Thr Asn Asp His Leu
515 520 525
Val Ala Leu Ala Cys Leu Gly Gly Arg Pro Ala Met Asp Ala Val Lys
530 535 540
Lys Gly Leu Pro His Ala Pro Glu Leu Ile Arg Arg Val Asn Arg Arg
545 550 555 560
Ile Gly Glu Arg Thr Ser His Arg Val Ala Ile Ser Arg Ser Arg Arg
565 570 575
Glu Ser Ile Asn Pro Trp Ile Leu Thr Gly Phe Ala Asp Ala Glu Gly
580 585 590
Ser Phe Gln Leu Glu Ile Arg Asn Val Asn Pro Asn Ile Pro Arg Tyr
595 600 605
Arg Thr Arg Leu Arg Phe Glu Ile Asp Leu His Asn Lys Asp Lys Ser
610 615 620
Ile Leu Glu Asp Ile Gln Ser Thr Trp Lys Val Gly Lys Ile Tyr Asn
625 630 635 640
Gln Gly Asp Ser Tyr Val Lys Leu Arg Val Thr Arg Phe Glu Asp Leu
645 650 655
Lys Val Ile Ile Asp His Phe Glu Lys Tyr Pro Leu Ile Thr Gln Lys
660 665 670
Leu Gly Asp Tyr Lys Leu Phe Lys Gln Ala Phe Ser Val Met Glu Asn
675 680 685
Lys Glu His Leu Lys Glu Asn Gly Ile Lys Glu Leu Val Arg Ile Lys
690 695 700
Ala Lys Met Asn Trp Gly Leu Asn Asp Glu Leu Lys Lys Ala Phe Pro
705 710 715 720
Glu Asn Ile Ser Lys Glu Arg Pro Leu Ile Asn Lys Asn Ile Pro Asn
725 730 735
Leu Lys Trp Leu Ala Gly Phe Thr Ser Gly Asp Gly His Phe Gly Val
740 745 750
Ile Leu Ala Lys Arg Arg Pro Ala Ser Pro Val Gln Val Arg Leu Arg
755 760 765
Phe Met Ile Gly Gln His Ile Lys Asp Lys Asn Leu Met Asn Ser Leu
770 775 780
Ile Thr Tyr Leu Gly Cys Gly Arg Ile Arg Glu Lys Asn Lys Ser Glu
785 790 795 800
Lys Ser Trp Leu Glu Phe Glu Val Thr Lys Phe Ser Asp Ile Asn Asp
805 810 815
Lys Ile Ile Pro Val Phe Gln Glu Asn Thr Leu Ile Gly Val Lys Leu
820 825 830
Glu Asp Phe Glu Asp Trp Cys Lys Val Ala Lys Leu Ile Glu Glu Lys
835 840 845
Lys His Leu Thr Glu Ser Gly Leu Asp Glu Ile Lys Lys Ile Lys Leu
850 855 860
Asn Met Asn Lys Gly Arg
865 870
<210>20
<211>841
<212>PRT
<213>Искусственная последовательность
<220>
<223>Синтезированный megaTAL PD-1.ile3.exon1_RD1 B1
<400>20
Met Gly Ser Ala Pro Pro Lys Lys Lys Arg Lys Val Val Asp Leu Arg
1 5 10 15
Thr Leu Gly Tyr Ser Gln Gln Gln Gln Glu Lys Ile Lys Pro Lys Val
20 25 30
Arg Ser Thr Val Ala Gln His His Glu Ala Leu Val Gly His Gly Phe
35 40 45
Thr His Ala His Ile Val Ala Leu Ser Gln His Pro Ala Ala Leu Gly
50 55 60
Thr Val Ala Val Thr Tyr Gln His Ile Ile Thr Ala Leu Pro Glu Ala
65 70 75 80
Thr His Glu Asp Ile Val Gly Val Gly Lys Gln Trp Ser Gly Ala Arg
85 90 95
Ala Leu Glu Ala Leu Leu Thr Asp Ala Gly Glu Leu Arg Gly Pro Pro
100 105 110
Leu Gln Leu Asp Thr Gly Gln Leu Val Lys Ile Ala Lys Arg Gly Gly
115 120 125
Val Thr Ala Met Glu Ala Val His Ala Ser Arg Asn Ala Leu Thr Gly
130 135 140
Ala Pro Leu Asn Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn
145 150 155 160
Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
165 170 175
Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala
180 185 190
Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
195 200 205
Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala
210 215 220
Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
225 230 235 240
Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val
245 250 255
Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val
260 265 270
Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp
275 280 285
Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu
290 295 300
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr
305 310 315 320
Pro Asp Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala
325 330 335
Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly
340 345 350
Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys
355 360 365
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp
370 375 380
His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser His Asp Gly
385 390 395 400
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
405 410 415
Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn
420 425 430
Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
435 440 445
Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala
450 455 460
Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Ser Ile Val Ala Gln Leu
465 470 475 480
Ser Arg Pro Asp Pro Ala Leu Ala Ala Leu Thr Asn Asp His Leu Val
485 490 495
Ala Leu Ala Cys Leu Gly Gly Arg Pro Ala Met Asp Ala Val Lys Lys
500 505 510
Gly Leu Pro His Ala Pro Glu Leu Ile Arg Arg Val Asn Arg Arg Ile
515 520 525
Gly Glu Arg Thr Ser His Arg Val Ala Ile Ser Arg Val Gly Gly Ser
530 535 540
Ser Arg Arg Glu Ser Ile Asn Pro Trp Ile Leu Thr Gly Phe Ala Asp
545 550 555 560
Ala Glu Gly Ser Phe Gly Leu Ser Ile Leu Asn Arg Asn Arg Gly Thr
565 570 575
Ala Arg Tyr His Thr Arg Leu Ser Phe Ala Ile Val Leu His Asn Lys
580 585 590
Asp Lys Ser Ile Leu Glu Asn Ile Gln Ser Thr Trp Lys Val Gly Ile
595 600 605
Ile Thr Asn Asp Gly Asp Arg Tyr Val Arg Leu Arg Val Thr Arg Phe
610 615 620
Glu Asp Leu Lys Val Ile Ile Asp His Phe Glu Lys Tyr Pro Leu Val
625 630 635 640
Thr Gln Lys Leu Gly Asp Tyr Lys Leu Phe Lys Gln Ala Phe Ser Val
645 650 655
Met Glu Asn Lys Glu His Leu Lys Glu Asn Gly Ile Lys Glu Leu Val
660 665 670
Arg Ile Lys Ala Lys Met Asn Trp Gly Leu Asn Asp Glu Leu Lys Lys
675 680 685
Ala Phe Pro Glu Asn Ile Ser Lys Glu Arg Pro Leu Ile Asn Lys Asn
690 695 700
Ile Pro Asn Phe Lys Trp Leu Ala Gly Phe Thr Ser Gly Asp Gly Ser
705 710 715 720
Phe Phe Val Arg Leu Arg Lys Ser Asn Val Asn Ala Arg Val Arg Val
725 730 735
Gln Leu Val Phe Glu Ile Ser Gln His Ile Arg Asp Lys Asn Leu Met
740 745 750
Asn Ser Leu Ile Thr Tyr Leu Gly Cys Gly His Ile Tyr Glu Gly Asn
755 760 765
Lys Ser Glu Arg Ser Trp Leu Gln Phe Arg Val Glu Lys Phe Ser Asp
770 775 780
Ile Asn Asp Lys Ile Ile Pro Val Phe Gln Glu Asn Thr Leu Ile Gly
785 790 795 800
Val Lys Leu Glu Asp Phe Glu Asp Trp Cys Lys Val Ala Lys Leu Ile
805 810 815
Glu Glu Lys Lys His Leu Thr Glu Ser Gly Leu Asp Glu Ile Lys Lys
820 825 830
Ile Lys Leu Asn Met Asn Lys Gly Arg
835 840
<210>21
<211>841
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Синтезированный megaTAL PD-1.ile3.exon1_RD2_B1H8
<400>21
Met Gly Ser Ala Pro Pro Lys Lys Lys Arg Lys Val Val Asp Leu Arg
1 5 10 15
Thr Leu Gly Tyr Ser Gln Gln Gln Gln Glu Lys Ile Lys Pro Lys Val
20 25 30
Arg Ser Thr Val Ala Gln His His Glu Ala Leu Val Gly His Gly Phe
35 40 45
Thr His Ala His Ile Val Ala Leu Ser Gln His Pro Ala Ala Leu Gly
50 55 60
Thr Val Ala Val Thr Tyr Gln His Ile Ile Thr Ala Leu Pro Glu Ala
65 70 75 80
Thr His Glu Asp Ile Val Gly Val Gly Lys Gln Trp Ser Gly Ala Arg
85 90 95
Ala Leu Glu Ala Leu Leu Thr Asp Ala Gly Glu Leu Arg Gly Pro Pro
100 105 110
Leu Gln Leu Asp Thr Gly Gln Leu Val Lys Ile Ala Lys Arg Gly Gly
115 120 125
Val Thr Ala Met Glu Ala Val His Ala Ser Arg Asn Ala Leu Thr Gly
130 135 140
Ala Pro Leu Asn Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn
145 150 155 160
Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
165 170 175
Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala
180 185 190
Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
195 200 205
Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala
210 215 220
Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
225 230 235 240
Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val
245 250 255
Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val
260 265 270
Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp
275 280 285
Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu
290 295 300
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr
305 310 315 320
Pro Asp Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala
325 330 335
Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly
340 345 350
Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys
355 360 365
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp
370 375 380
His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser His Asp Gly
385 390 395 400
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
405 410 415
Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn
420 425 430
Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
435 440 445
Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala
450 455 460
Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Ser Ile Val Ala Gln Leu
465 470 475 480
Ser Arg Pro Asp Pro Ala Leu Ala Ala Leu Thr Asn Asp His Leu Val
485 490 495
Ala Leu Ala Cys Leu Gly Gly Arg Pro Ala Met Asp Ala Val Lys Lys
500 505 510
Gly Leu Pro His Ala Pro Glu Leu Ile Arg Arg Val Asn Arg Arg Ile
515 520 525
Gly Glu Arg Thr Ser His Arg Val Ala Ile Ser Arg Val Gly Gly Ser
530 535 540
Ser Arg Arg Glu Ser Ile Asn Pro Trp Ile Leu Thr Gly Phe Ala Asp
545 550 555 560
Ala Glu Gly Ser Phe Gly Leu Ser Ile Leu Asn Arg Asn Arg Gly Thr
565 570 575
Ala Arg Tyr His Thr Arg Leu Ser Phe Thr Ile Val Leu His Asn Lys
580 585 590
Asp Lys Ser Ile Leu Glu Asn Ile Gln Ser Thr Trp Lys Val Gly Ile
595 600 605
Ile Thr Asn Asp Gly Asp Arg Tyr Val Arg Leu Cys Val Thr Arg Phe
610 615 620
Glu Asp Leu Lys Val Ile Ile Asp His Phe Glu Lys Tyr Pro Leu Val
625 630 635 640
Thr Gln Lys Leu Gly Asp Tyr Lys Leu Phe Lys Gln Ala Phe Ser Val
645 650 655
Met Glu Asn Lys Glu His Leu Lys Glu Asn Gly Ile Lys Glu Leu Ala
660 665 670
Arg Ile Lys Ala Lys Met Asn Trp Gly Leu Asn Asp Glu Leu Lys Lys
675 680 685
Ala Phe Pro Glu Asn Ile Gly Lys Glu Arg Pro Leu Ile Asn Lys Asn
690 695 700
Ile Pro Asn Phe Lys Trp Leu Ala Gly Phe Thr Ser Gly Asp Gly Ser
705 710 715 720
Phe Phe Val Arg Leu Arg Lys Ser Asn Val Asn Ala Arg Val Arg Val
725 730 735
Gln Leu Val Phe Glu Ile Ser Gln His Ile Arg Asp Lys Asn Leu Met
740 745 750
Asn Ser Leu Ile Thr Tyr Leu Gly Cys Gly His Ile Tyr Glu Gly Asn
755 760 765
Lys Ser Glu Arg Ser Trp Leu Gln Phe Arg Val Glu Lys Phe Ser Asp
770 775 780
Ile Asn Asp Lys Ile Ile Pro Val Phe Gln Glu Asn Thr Leu Ile Gly
785 790 795 800
Val Lys Leu Glu Asp Phe Glu Asp Trp Cys Lys Val Ala Lys Leu Ile
805 810 815
Glu Glu Lys Lys His Leu Thr Glu Ser Gly Leu Asp Glu Ile Lys Lys
820 825 830
Ile Lys Leu Asn Met Asn Lys Gly Arg
835 840
<210>22
<211>909
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Синтезированный megaTAL PD-1.IgV.exon2_RD1_G5
<400>22
Met Gly Ser Ala Pro Pro Lys Lys Lys Arg Lys Val Val Asp Leu Arg
1 5 10 15
Thr Leu Gly Tyr Ser Gln Gln Gln Gln Glu Lys Ile Lys Pro Lys Val
20 25 30
Arg Ser Thr Val Ala Gln His His Glu Ala Leu Val Gly His Gly Phe
35 40 45
Thr His Ala His Ile Val Ala Leu Ser Gln His Pro Ala Ala Leu Gly
50 55 60
Thr Val Ala Val Thr Tyr Gln His Ile Ile Thr Ala Leu Pro Glu Ala
65 70 75 80
Thr His Glu Asp Ile Val Gly Val Gly Lys Gln Trp Ser Gly Ala Arg
85 90 95
Ala Leu Glu Ala Leu Leu Thr Asp Ala Gly Glu Leu Arg Gly Pro Pro
100 105 110
Leu Gln Leu Asp Thr Gly Gln Leu Val Lys Ile Ala Lys Arg Gly Gly
115 120 125
Val Thr Ala Met Glu Ala Val His Ala Ser Arg Asn Ala Leu Thr Gly
130 135 140
Ala Pro Leu Asn Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn
145 150 155 160
Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
165 170 175
Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala
180 185 190
Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
195 200 205
Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala
210 215 220
Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
225 230 235 240
Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val
245 250 255
Val Ala Ile Ala Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu Thr Val
260 265 270
Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp
275 280 285
Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu
290 295 300
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr
305 310 315 320
Pro Asp Gln Val Val Ala Ile Ala Ser Asn Ile Gly Gly Lys Gln Ala
325 330 335
Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly
340 345 350
Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys
355 360 365
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp
370 375 380
His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn Ile Gly
385 390 395 400
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
405 410 415
Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn
420 425 430
Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
435 440 445
Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala
450 455 460
Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
465 470 475 480
Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala
485 490 495
Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
500 505 510
Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val
515 520 525
Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Ser Ile
530 535 540
Val Ala Gln Leu Ser Arg Pro Asp Pro Ala Leu Ala Ala Leu Thr Asn
545 550 555 560
Asp His Leu Val Ala Leu Ala Cys Leu Gly Gly Arg Pro Ala Met Asp
565 570 575
Ala Val Lys Lys Gly Leu Pro His Ala Pro Glu Leu Ile Arg Arg Val
580 585 590
Asn Arg Arg Ile Gly Glu Arg Thr Ser His Arg Val Ala Ile Ser Arg
595 600 605
Val Gly Gly Ser Ser Arg Arg Glu Ser Ile Asn Pro Trp Ile Leu Thr
610 615 620
Gly Phe Ala Asp Ala Glu Gly Ser Phe Gln Leu Tyr Ile Ser Asn Val
625 630 635 640
Asn Asn Asn Arg Ser Arg Tyr Arg Ala Arg Leu Arg Phe Ala Ile Glu
645 650 655
Leu His Asn Lys Asp Lys Ser Ile Leu Glu Asn Ile Gln Ser Thr Trp
660 665 670
Lys Val Gly Val Ile Asn Asn Ile Gly Asp Thr Ser Val Arg Leu Ser
675 680 685
Val Gly Arg Phe Glu Asp Leu Lys Val Ile Ile Asp His Phe Glu Lys
690 695 700
Tyr Pro Leu Ile Thr Gln Lys Leu Gly Asp Tyr Lys Leu Phe Lys Gln
705 710 715 720
Ala Phe Ser Val Met Glu Asn Lys Glu His Leu Lys Glu Asn Gly Ile
725 730 735
Lys Glu Leu Val Arg Ile Lys Ala Lys Met Asn Trp Gly Leu Asn Asp
740 745 750
Glu Leu Lys Lys Ala Phe Pro Glu Asn Ile Ser Lys Glu Arg Pro Leu
755 760 765
Ile Asn Lys Asn Ile Pro Asn Leu Lys Trp Leu Ala Gly Phe Thr Ser
770 775 780
Gly Asp Gly Asn Phe Tyr Val His Leu Lys Lys Ser Gly Arg Thr Thr
785 790 795 800
Arg Val Tyr Val Gln Leu Arg Phe Ser Ile Ala Gln His Ile Arg Asp
805 810 815
Lys Asn Leu Met Asn Ser Leu Ile Thr Tyr Leu Gly Cys Gly Tyr Ile
820 825 830
Asn Glu Trp Asn Ala Ser Glu Arg Ser Ala Leu Glu Phe Arg Val Thr
835 840 845
Lys Phe Ser Asp Ile Asn Asp Lys Ile Ile Pro Val Phe Gln Glu Asn
850 855 860
Thr Leu Ile Gly Val Lys Leu Glu Asp Phe Glu Asp Trp Cys Lys Val
865 870 875 880
Ala Lys Leu Ile Glu Glu Lys Lys His Leu Thr Glu Ser Gly Leu Asp
885 890 895
Glu Ile Lys Lys Ile Lys Leu Asn Met Asn Lys Gly Arg
900 905
<210>23
<211>909
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Синтезированный megaTAL PD-1.IgV.exon2_RD1_PS3
<400>23
Met Gly Ser Ala Pro Pro Lys Lys Lys Arg Lys Val Val Asp Leu Arg
1 5 10 15
Thr Leu Gly Tyr Ser Gln Gln Gln Gln Glu Lys Ile Lys Pro Lys Val
20 25 30
Arg Ser Thr Val Ala Gln His His Glu Ala Leu Val Gly His Gly Phe
35 40 45
Thr His Ala His Ile Val Ala Leu Ser Gln His Pro Ala Ala Leu Gly
50 55 60
Thr Val Ala Val Thr Tyr Gln His Ile Ile Thr Ala Leu Pro Glu Ala
65 70 75 80
Thr His Glu Asp Ile Val Gly Val Gly Lys Gln Trp Ser Gly Ala Arg
85 90 95
Ala Leu Glu Ala Leu Leu Thr Asp Ala Gly Glu Leu Arg Gly Pro Pro
100 105 110
Leu Gln Leu Asp Thr Gly Gln Leu Val Lys Ile Ala Lys Arg Gly Gly
115 120 125
Val Thr Ala Met Glu Ala Val His Ala Ser Arg Asn Ala Leu Thr Gly
130 135 140
Ala Pro Leu Asn Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn
145 150 155 160
Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
165 170 175
Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala
180 185 190
Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
195 200 205
Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala
210 215 220
Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
225 230 235 240
Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val
245 250 255
Val Ala Ile Ala Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu Thr Val
260 265 270
Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp
275 280 285
Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu
290 295 300
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr
305 310 315 320
Pro Asp Gln Val Val Ala Ile Ala Ser Asn Ile Gly Gly Lys Gln Ala
325 330 335
Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly
340 345 350
Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys
355 360 365
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp
370 375 380
His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn Ile Gly
385 390 395 400
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
405 410 415
Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn
420 425 430
Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
435 440 445
Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala
450 455 460
Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
465 470 475 480
Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala
485 490 495
Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
500 505 510
Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val
515 520 525
Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Ser Ile
530 535 540
Val Ala Gln Leu Ser Arg Pro Asp Pro Ala Leu Ala Ala Leu Thr Asn
545 550 555 560
Asp His Leu Val Ala Leu Ala Cys Leu Gly Gly Arg Pro Ala Met Asp
565 570 575
Ala Val Lys Lys Gly Leu Pro His Ala Pro Glu Leu Ile Arg Arg Val
580 585 590
Asn Arg Arg Ile Gly Glu Arg Thr Ser His Arg Val Ala Ile Ser Arg
595 600 605
Val Gly Gly Ser Ser Arg Arg Glu Ser Ile Asn Pro Trp Ile Leu Thr
610 615 620
Gly Phe Ala Asp Ala Glu Gly Cys Phe Leu Leu His Ile Arg Asn Leu
625 630 635 640
Asn Arg Thr Ser Thr Lys Tyr Arg Thr Arg Leu Ser Phe Glu Ile Glu
645 650 655
Leu His Asn Lys Asp Lys Ser Ile Leu Glu Asn Ile Gln Ser Thr Trp
660 665 670
Lys Val Gly Ile Ile Asn Asn Ile Gly Asn Arg Arg Val Arg Leu Ser
675 680 685
Val Arg Arg Phe Glu Asp Leu Lys Val Ile Ile Asp His Phe Glu Lys
690 695 700
Tyr Pro Leu Ile Thr Gln Lys Leu Gly Asp Tyr Lys Leu Phe Lys Gln
705 710 715 720
Ala Phe Ser Leu Met Glu Asn Lys Glu His Leu Lys Glu Asn Gly Ile
725 730 735
Lys Glu Leu Val Arg Ile Lys Ala Lys Met Asn Trp Gly Leu Asn Asp
740 745 750
Glu Leu Lys Lys Ala Phe Pro Glu Asn Ile Ser Lys Glu Arg Pro Leu
755 760 765
Ile Asn Lys Asn Ile Pro Asn Leu Lys Trp Leu Ala Gly Phe Thr Ser
770 775 780
Gly Asp Gly Asn Phe Tyr Val His Leu Lys Lys Ser Gly Arg Thr Thr
785 790 795 800
Arg Val Tyr Val Gln Leu Arg Phe Ser Ile Ser Gln His Ile Arg Asp
805 810 815
Lys Asn Leu Met Asn Ser Leu Ile Thr Tyr Leu Gly Cys Gly Tyr Ile
820 825 830
Thr Glu Ser Asn Pro Ser Glu Arg Ser Asp Leu Glu Phe Arg Val Thr
835 840 845
Lys Phe Ser Asp Ile Asn Asp Lys Ile Ile Pro Val Phe Gln Glu Asn
850 855 860
Thr Leu Ile Gly Val Lys Leu Glu Asp Phe Glu Asp Trp Cys Lys Val
865 870 875 880
Ala Lys Leu Ile Glu Glu Lys Lys His Leu Thr Glu Ser Gly Leu Asp
885 890 895
Glu Ile Lys Lys Ile Lys Leu Asn Met Asn Lys Gly Arg
900 905
<210>24
<211>236
<212>БЕЛОК
<213>Mus musculus
<400>24
Met Ser Glu Pro Pro Arg Ala Glu Thr Phe Val Phe Leu Asp Leu Glu
1 5 10 15
Ala Thr Gly Leu Pro Asn Met Asp Pro Glu Ile Ala Glu Ile Ser Leu
20 25 30
Phe Ala Val His Arg Ser Ser Leu Glu Asn Pro Glu Arg Asp Asp Ser
35 40 45
Gly Ser Leu Val Leu Pro Arg Val Leu Asp Lys Leu Thr Leu Cys Met
50 55 60
Cys Pro Glu Arg Pro Phe Thr Ala Lys Ala Ser Glu Ile Thr Gly Leu
65 70 75 80
Ser Ser Glu Ser Leu Met His Cys Gly Lys Ala Gly Phe Asn Gly Ala
85 90 95
Val Val Arg Thr Leu Gln Gly Phe Leu Ser Arg Gln Glu Gly Pro Ile
100 105 110
Cys Leu Val Ala His Asn Gly Phe Asp Tyr Asp Phe Pro Leu Leu Cys
115 120 125
Thr Gly Leu Gln Arg Leu Gly Ala His Leu Pro Gln Asp Thr Val Cys
130 135 140
Leu Asp Thr Leu Pro Ala Leu Arg Gly Leu Asp Arg Ala His Ser His
145 150 155 160
Gly Thr Arg Ala Gln Gly Arg Lys Ser Tyr Ser Leu Ala Ser Leu Phe
165 170 175
His Arg Tyr Phe Gln Ala Glu Pro Ser Ala Ala His Ser Ala Glu Gly
180 185 190
Asp Val His Thr Leu Leu Leu Ile Phe Leu His Arg Ala Pro Glu Leu
195 200 205
Leu Ala Trp Ala Asp Glu Gln Ala Arg Ser Trp Ala His Ile Glu Pro
210 215 220
Met Tyr Val Pro Pro Asp Gly Pro Ser Leu Glu Ala
225 230 235
<210>25
<211>22
<212>ДНК
<213>Homo sapiens
<400>25
aatggtggca tactccgtct gc 22
<210>26
<211>11
<212>ДНК
<213>Homo sapiens
<400>26
tccgctagga a 11
<210>27
<211>37
<212>ДНК
<213>Homo sapiens
<400>27
tccgctagga aagacaatgg tggcatactc cgtctgc 37
<210>28
<211>22
<212>ДНК
<213>Homo sapiens
<400>28
aatggtggca tacaaccttt ta 22
<210>29
<211>22
<212>ДНК
<213>Homo sapiens
<400>29
tttccactta tactccgtct gc 22
<210>30
<211>22
<212>ДНК
<213>Homo sapiens
<400>30
ggcatgcaga tcccacaggc gc 22
<210>31
<211>14
<212>ДНК
<213>Homo sapiens
<400>31
ggtggggctg ctcc 14
<210>32
<211>37
<212>ДНК
<213>Homo sapiens
<400>32
ggtggggctg ctccaggcat gcagatccca caggcgc 37
<210>33
<211>22
<212>ДНК
<213>Homo sapiens
<400>33
ggcatgcaga tccaaccttt ta 22
<210>34
<211>22
<212>ДНК
<213>Homo sapiens
<400>34
tttccactta tcccacaggc gc 22
<210>35
<211>22
<212>ДНК
<213>Homo sapiens
<400>35
acgggcgtga cttccacatg ag 22
<210>36
<211>12
<212>ДНК
<213>Homo sapiens
<400>36
gtcacacaac tg 12
<210>37
<211>38
<212>ДНК
<213>Homo sapiens
<400>37
gtcacacaac tgcccaacgg gcgtgacttc cacatgag 38
<210>38
<211>22
<212>ДНК
<213>Homo sapiens
<400>38
acgggcgtga cttaaccttt ta 22
<210>39
<211>22
<212>ДНК
<213>Homo sapiens
<400>39
tttccactta cttccacatg ag 22
<210>40
<211>2650
<212>РНК
<213>Искусственная последовательность
<220>
<223>Синтезированная мРНК megaTAL RD5_CV23MK экзона 5 PD-1
<400>40
augggauccg ccccccccaa gaagaagcgc aagguggugg accugagaac ccugggguac 60
agccagcagc aacaggagaa gaucaaaccg aaggugcgca gcacgguggc ccagcaccau 120
gaagcccucg ugggucaugg auucacgcac gcccacaucg uggcccuguc gcagcauccg 180
gccgcccugg gcaccguggc ggugaccuac cagcacauca ucaccgcccu gcccgaagcc 240
acccacgagg acaucguggg agugggcaag cagugguccg gagcacgcgc ccuggaggcc 300
cugcugaccg acgccgggga acugcgcggc ccgccucugc agcuggauac cggccaacug 360
gucaagaucg ccaagagagg cggagugacc gcgauggagg ccguccacgc cucccggaac 420
gcucugaccg gugccccgcu caaucugacu ccggaccagg ugguggcuau cgccagcaac 480
ggaggaggaa aacaggcccu cgaaacagug cagaggcugc ugccuguccu uugucaagau 540
cacgggcuga cucccgacca ggucguggcc auugccagcc acgacggcgg caagcaggcu 600
uuggagacug ugcagcggcu ccugccagug cugugccaag aucacggucu gaccccagau 660
caggucgucg ccauugcuuc caacggaggc aaacaagcgc uggaaacggu ccaacgccug 720
cugccggugc uuugucagga ucacggccug accccugauc aggugguggc caucgcgucc 780
aauaacgggg ggaagcaggc acucgagacu guccagaggc uccugccugu gcucugccag 840
gaccacgggc ucacacccga ucaggucguc gcuaucgcgu cgcacgacgg uggaaagcag 900
gcccuggaaa ccgugcagcg ccuguugccg gugcuguguc aggaccaugg ccuuacuccg 960
gaucaggucg ucgcgaucgc aucuaauggu ggaggaaagc aggcccugga gacaguccag 1020
cgccugcucc cgguguugug ccaagaccau ggucuuaccc cugaccaggu ggucgcuauu 1080
gccucgaaca ucggcggaaa gcaagcccug gaaaccgugc agcgacuucu gccgguccug 1140
ugccaggauc auggauugac cccagaccag gugguggcga uugccagcaa caacggcggg 1200
aagcaagcgu uggaaaccgu ccagagacug cuuccugugc ugugccaaga ccacgguuug 1260
accccggacc aagucgucgc caucgcuucc aacaacggag ggaagcaggc acucgaaacu 1320
gugcaacggu ugcugcccgu gcucugucag gaucacggac ucaccccuga ucagguggug 1380
gccaucgcaa gcaacaucgg uggcaaacag gcucuggaaa cuguccaaag acugcugccc 1440
gugcuuugcc aggaccacgg acugacuccu gaccaagugg uggcaauugc cuccaacauc 1500
ggaggcaagc aagcgcucga auccaucgug gcgcagcuca gccggccaga ccccgcccug 1560
gccgcccuga cuaacgauca ccugguggcc cuggcgugcc ucggcggucg ccccgcuaug 1620
gacgcgguga agaaggggcu gccccacgcc cccgagcuca uucggcgggu gaaccgccgg 1680
aucggagaaa gaaccuccca ucggguggcc aucucgagau cacggcggga auccauuaac 1740
cccuggaucc ugacuggcuu ugccgacgcc gagggauccu uccagcucga aauccggaac 1800
gugaacccaa acaucccccg guaucgcacc agacugcggu ucgagaucga ccuucacaac 1860
aaggacaagu ccauucugga ggacauccag ucaaccugga aagugggaaa gaucuacaac 1920
cagggggacu cauacgugaa gcugcgggug acccgcuucg aagaucucaa agugaucauc 1980
gaccauuucg agaaguaccc ccugaucacu cagaagcuug gagacuacaa acuguucaag 2040
caggcauucu ccgugaugga gaacaaggag caccugaagg agaacgggau uaaggagcug 2100
gugcgaauua aggcgaaaau gaacugggga uugaacgaug agcugaagaa ggcguucccg 2160
gaaaacauuu ccaaggagcg cccgcucauc aacaagaaca ucccuaaucu gaaguggcuc 2220
gcgggguuua ccucuggcga cggccauuuc ggagugauuc ucgcaaagcg caggcccgcc 2280
agcccugugc aagugcggcu gcgguucaug aucggccagc acauuaagga caagaaccug 2340
augaacucgc ucaucaccua ccuuggaugc ggccggauuc gcgaaaagaa caagagcgag 2400
aaguccuggc uggaauuuga agugaccaag uucagcgaca ucaacgacaa gaucauuccg 2460
guguuccagg aaaacacccu gauuggcgug aagcuggagg acuucgagga uuggugcaag 2520
guggccaagc ucaucgaaga aaagaagcac cugacugaau ccggcuugga cgagaucaag 2580
aagauuaagc ugaauaugaa caagggaagg ugauagcgcg cuagccguac ggaaaaaaaa 2640
aaaaaaaaaa 2650
<210>41
<211>2526
<212>РНК
<213>Искусственная последовательность
<220>
<223>Синтезированная мРНК megaTAL RD2_B1H8 экзона 1 PD-1
<400>41
augggaagcg ccccgccgaa gaagaagcgc aagguggugg aucugagaac ccugggauac 60
agccagcagc agcaggagaa gaucaagccg aagguccggu cuaccguggc ccagcaccau 120
gaggcccuug ugggccacgg cuucacacau gcacacaucg ucgcccuguc gcagcauccc 180
gccgcccugg ggaccguggc cgugaccuau caacacauca uuaccgcccu gccggaggcc 240
acccacgagg acaucguggg uguggggaag caguggagcg gagccagggc acucgaagcc 300
cuccucacug acgcuggaga acugcgcgga ccgccucucc agcuggacac cggacagcug 360
gugaaaaucg ccaagcgggg aggagugacc gccauggaag ccgugcacgc cucgaggaac 420
gcgcugacug gcgccccucu gaaccugacc ccugaucagg ucguggcuau cgccucaaac 480
aacgggggua agcaggcgcu ggagacagug caacgacuuc ugccagugcu uugucaggac 540
cauggucuga cccccgacca ggucgucgcc auugcaucca acaauggugg caagcaggca 600
cuggagacug uccagaggcu gcucccggug cugugccagg accacgggcu caccccggac 660
caaguggucg ccaucgccuc caacggagga ggaaaacaag cucuggagac ugugcaacgc 720
cugcugccug uguugugcca agaccacgga cugacgcccg aucagguggu ggcgaucgca 780
ucgaacaacg gaggaaagca agcgcuggaa accgugcagc gccuccugcc cguccucugc 840
caggaucacg gccugacucc ggaccaggug gucgcgaucg ccagcaauaa cggggggaag 900
caagcccucg agacugugca gcgguugcug cccgugcucu gccaagauca uggccuuacc 960
ccagaccaag ucguggccau ugcuuccaac aacgguggca aacaggcgcu cgaaaccguc 1020
cagcggcugu ugcccgugcu uugccaggau cacggacuca ccccugauca ggugguggca 1080
auugcgucca acaacggugg aaagcaggcc cuggaaacgg ugcagcggcu gcuuccgguc 1140
cugugucagg aucaugggcu gacucccgac caggucgucg ccauugcauc ccacgauggg 1200
gguaaacagg cccucgaaac agugcagaga cuccugccag uccugugcca agaccacgga 1260
cuuaccccgg aucagguggu ggccauagcc ucgaacggcg gcgggaaaca ggcucuggaa 1320
acugugcaaa gacuccuccc gguguugugu caagaccaug gacugacccc agaucaggug 1380
guggcuauug ccucuaacaa cggcggcaag caagcacucg aaagcaucgu ggcccaguug 1440
ucacgccccg accccgcacu ggcugcccug acgaaugacc aucugguggc gcuggccugc 1500
cugggaggga ggccagcgau ggaugcggug aagaagggac ugccccaugc uccggagcug 1560
auucggagag ugaauaggcg caucggagag agaacuucac aucggguggc cauuucuaga 1620
gugggcggca gcucccggcg cgaguccauu aaccccugga uccugaccgg cuuugccgac 1680
gccgaagggu ccuucggccu cucgauccug aaccggaacc gggguaccgc ucgguaccac 1740
accagacugu ccuucaccau cgugcugcac aacaaggaca agagcauccu cgaaaacauu 1800
cagucaacgu ggaagguggg aauuauuacu aacgacggcg acagauacgu gcgccugugc 1860
gugacccggu uugaggaccu gaaggucauu aucgaccacu ucgagaagua cccccucgug 1920
acucagaagc ugggagacua caagcuguuc aagcaggcgu ucucggugau ggaaaacaag 1980
gagcaccuga aggagaacgg caucaaggag cucgcccgga ucaaggccaa gaugaacugg 2040
ggccugaaug augaacucaa gaaggcguuc ccugaaaaca ucgguaaaga acggccccug 2100
aucaacaaga acaucccgaa cuucaagugg cuugccggau ucaccuccgg cgacggaucc 2160
uucuucgucc ggcugcgcaa guccaacgug aacgcgagag ugcgggugca auuggucuuu 2220
gaaaucucac agcacaucag ggacaagaau uugaugaacu cccucaucac cuaccugggu 2280
ugcggacaca ucuacgaagg caauaagucg gagcggagcu ggcugcaguu ucgcguggaa 2340
aaguucuccg acauuaacga caagaucauc ccaguguucc aggaaaacac ucugauuggc 2400
gugaagcuug aggauuucga ggacuggugc aagguggcca agcugauuga agagaagaag 2460
caccugaccg aguccggccu ggacgaaauc aagaaaauca agcugaacau gaacaaggga 2520
cgguga 2526
<210>42
<211>2730
<212>РНК
<213>Искусственная последовательность
<220>
<223>Синтезированная мРНК megaTAL RD1_G5 экзона 2 PD-1
<400>42
augggaagcg ccccuccgaa gaagaagcgc aaggucgugg accucagaac ccuggguuac 60
ucccagcagc agcaggagaa aauuaagccg aaggugcgcu ccaccguggc ucaacaccac 120
gaggcccucg ugggccaugg auucacucac gcccauaucg uggcccuguc ccagcacccg 180
gccgcccugg gcaccguggc ggugaccuac cagcacauca ucaccgcgcu gccugaagcc 240
acccacgagg acaucgucgg uguggguaag cagugguccg gagccagagc ccuggaggcu 300
cugcugaccg acgccggaga acucagaggc ccgccucugc agcuggacac cggacagcug 360
gugaagauag ccaagagagg cggcgugacc gccauggaag ccgugcaugc gucccgcaac 420
gcacugaccg gggccccccu gaaccugacu ccagaccaag ugguggcuau cgccagcaac 480
aauggaggaa agcaagccuu ggaaaccgug cagcggcugc ucccgguccu uugccaagac 540
cacggccuga cacccgauca ggugguggca aucgcaucga auggcggcgg gaagcaggcc 600
cucgagacug ugcagaggcu ccugccugug cugugccagg accaugggcu gaccccagac 660
cagguggucg caaucgccuc gcacgacgga ggcaagcaag cccuggaaac uguccagcgc 720
cugcucccug uccuguguca agaucauggg cucacuccug aucaggucgu cgccaucgcc 780
ucgaacauug guggcaagca ggcgcucgaa accguccagc gguugcugcc agugcuuugc 840
caggaccaug gucugacccc cgaucaagug gucgcgauug ccucacacga uggcgguaag 900
caggcuuugg aaaccgugca acggcuguug ccuguccucu gccaggacca cggcuugacu 960
cccgaccagg ugguggccau agccucaaac aucggaggga aacaagcccu cgaaaccguc 1020
cagaggcugc ugccgguguu gugccaggau cacggauuga ccccagacca gguggucgcc 1080
auugcuuccc acgauggggg aaagcaggcc cuggagacug ugcagcgccu ccuucccguc 1140
cugugucagg aucauggacu uacccccgac caagucgugg cgauugcuuc caauaucgga 1200
ggcaaacagg cccuugaaac agugcagcgc cuguugccgg ugcucugcca agaucacgga 1260
cugaccccug accagguggu ggcgaucgcg ucaaauaucg gcggcaagca ggcacuggag 1320
acaguccaga gacuccugcc gguccucugc caggaccacg gucuuacucc ugaccaagug 1380
guggcuaucg caucccauga uggugggaaa caggcucuug aaacugugca acgccugcug 1440
cccgugcugu gccaagacca cggacugacu ccggaccagg ucguggccau cgcuucaaac 1500
ggagggggaa aacaggcacu ugaaacggug cagagacugc ugccuguccu uugucaggac 1560
cacggguuga ccccggacca ggucguggcu auugccucga acaacggggg gaagcaagcg 1620
cucgagucca uuguggccca gcugagccgg ccugaucccg cacuggccgc gcugaccaac 1680
gaucaccugg uggcccucgc cugucugggc ggacggccgg ccauggacgc cgugaagaag 1740
ggacugccgc acgcgcccga gcugauccgc cgcgugaaca ggcggauugg agaacgcacc 1800
ucccaccggg uggccaucuc gagaguggga ggauccucua gacgggaguc cauuaacccg 1860
uggauccuga cuggauucgc cgacgccgag ggguccuucc agcuguacau cuccaacgug 1920
aacaacaacc gcagcagaua cagggcccgg cugcgguucg cgaucgaacu gcacaauaag 1980
gacaagagca uccucgagaa cauucagucc acuuggaagg ugggcgugau uaacaacauc 2040
ggcgauacca gcgugcggcu cuccgugggg cgguucgaag aucucaaggu gaucaucgac 2100
cacuucgaga aguacccgcu gaucacccag aagcugggag acuacaagcu guucaagcaa 2160
gcuuucagcg ugauggaaaa caaggaacau cugaaagaga acgguaucaa ggaacuggug 2220
cggauuaagg ccaagaugaa cuggggucug aacgaugaac ugaagaaggc guucccggag 2280
aauaucagca aggagcgccc ccugauuaac aaaaacaucc caaaccucaa guggcucgcc 2340
ggcuuuacuu ccggagaugg uaacuucuac gugcaccuga agaaguccgg aagaaccacc 2400
cgggucuacg ugcagcugag guucuccauc gcccagcaca uccgggacaa gaacuugaug 2460
aacucccuga ucaccuaccu cgguugcgga uacaucaacg aauggaacgc cagcgagaga 2520
ucggcccugg aguucagggu gacuaaguuc uccgacauca acgacaagau uauccccgug 2580
uuucaggaaa auacccucau cggcgugaag cuggaggacu uugaggacug gugcaaggug 2640
gccaagcuga ucgaggaaaa gaaacaucug accgagagcg gccuggacga aaucaagaag 2700
auuaagcuga acaugaacaa gggacgauga 2730
<210>43
<211>711
<212>РНК
<213>Mus musculus
<400>43
augucugagc caccucgggc ugagaccuuu guauuccugg accuagaagc cacugggcuc 60
ccaaacaugg acccugagau ugcagagaua ucccuuuuug cuguucaccg cucuucccug 120
gagaacccag aacgggauga uucugguucc uuggugcugc cccguguucu ggacaagcuc 180
acacugugca ugugcccgga gcgccccuuu acugccaagg ccagugagau uacugguuug 240
agcagcgaaa gccugaugca cugcgggaag gcugguuuca auggcgcugu gguaaggaca 300
cugcagggcu uccuaagccg ccaggagggc cccaucugcc uuguggccca caauggcuuc 360
gauuaugacu ucccacugcu gugcacgggg cuacaacguc ugggugccca ucugccccaa 420
gacacugucu gccuggacac acugccugca uugcggggcc uggaccgugc ucacagccac 480
ggcaccaggg cucaaggccg caaaagcuac agccuggcca gucucuucca ccgcuacuuc 540
caggcugaac ccagugcugc ccauucagca gaaggugaug ugcacacccu gcuucugauc 600
uuccugcauc gugcuccuga gcugcucgcc ugggcagaug agcaggcccg cagcugggcu 660
cauauugagc ccauguacgu gccaccugau gguccaagcc ucgaagccug a 711
<210>44
<211>7601
<212>ДНК
<213>Искусственная последовательность
<220>
<223>Синтезированная конструкция Exon5_MND-BFP rAAV PD-1
<400>44
cagctgcgcg ctcgctcgct cactgaggcc gcccgggcaa agcccgggcg tcgggcgacc 60
tttggtcgcc cggcctcagt gagcgagcga gcgcgcagag agggagtggc caactccatc 120
actaggggtt ccttgtagtt aatgattaac ccgccatgct acttatctac gtagaattca 180
gtccagggct ctgtcctgca cctggggaat ggtgaccggc atctctgtcc tctagctctg 240
gaagcacccc agcccctcta gtctgccctc acccctgacc ctgaccctcc accctgaccc 300
cgtcctaacc cctgaccttt gtgcccttcc agagagaagg gcagaagtgc ccacagccca 360
ccccagcccc tcacccaggc cagccggcca gttccaaacc ctggtggttg gtgtcgtggg 420
cggcctgctg ggcagcctgg tgctgctagt ctgggtcctg gccgtcatct gctcccgggc 480
cgcacgaggt aacgtcatcc cagcccctcg gcctgccctg ccctaaccct gctggcggcc 540
ctcactcccg cctccccttc ctccaccctt ccctcacccc accccacctc cccccatctc 600
cccgccaggc taagtccctg atgaaggccc ctggactaag accccccacc taggagcacg 660
gctcagggtc ggcctggtga ccccaagtgt gtttctctgc agggacaata ggagccaggc 720
gcaccggcca gcccctggtg agtctcactc ttttcctgca tgatccactg tgccttcctt 780
cctgggtggg cagaggtgga aggacaggct gggaccacac ggcctgcagg actcacattc 840
tattatagcc aggaccccac ctccccagcc cccaggcagc aacctcaatc cctaaagcca 900
tgatctgggg ccccagccca cctgcggtct ccgggggtgc ccggcccatg tgtgtgcctg 960
cctgcggtct ccaggggtgc ctggcccacg cgtgtgcccg cctgcggtct ctgggggtgc 1020
ccggcccaca tatgtgcctg cctgcggtct ccaggtgtgc ccggcccatg cgtgtgccca 1080
cctgcgaggg cgtggggtgg gcttggtcat ttcttatctt acattggaga caggagagct 1140
tgaaaagtca cattttggaa tcctaaatct gcaagaatgc cagggacatt tcagaggggg 1200
acattgagcc agagaggagg ggtggtgtcc ccagatcaca cagagggcag tggtgggaca 1260
gctcagggta agcagctcat agtggggggc ccaggttcgg tgccggtact gcagccaggc 1320
tgtggagccg cgggcctcct tcctgcggtg ggccgtgggg ctgactccct ctccctttct 1380
cctcaaagaa ggaggacccc tcagccgtgc ctgtgttctc tgtggactat ggggagctgg 1440
atttccagtg gcgagagaag accccggagc cccccgtgcc ctgtgtccct gagcagacgg 1500
agtaatgcat gaacagagaa acaggagaat atgggccaaa caggatatct gtggtaagca 1560
gttcctgccc cggctcaggg ccaagaacag ttggaacagc agaatatggg ccaaacagga 1620
tatctgtggt aagcagttcc tgccccggct cagggccaag aacagatggt ccccagatgc 1680
ggtcccgccc tcagcagttt ctagagaacc atcagatgtt tccagggtgc cccaaggacc 1740
tgaaatgacc ctgtgcctta tttgaactaa ccaatcagtt cgcttctcgc ttctgttcgc 1800
gcgcttctgc tccccgagct ctatataagc agagctcgtt tagtgaaccg tcagatcgcc 1860
tggagacgcc atccacgctg ttttgacttc catagaagga tctcgaggcc accatggcta 1920
gcgagctgat taaggagaac atgcacatga agctgtacat ggagggcacc gtggacaacc 1980
atcacttcaa gtgcacatcc gagggcgaag gcaagcccta cgagggcacc cagaccatga 2040
gaatcaaggt ggtcgagggc ggccctctcc ccttcgcctt cgacatcctg gctactagct 2100
tcctctacgg cagcaagacc ttcatcaacc acacccaggg catccccgac ttcttcaagc 2160
agtccttccc tgagggcttc acatgggaga gagtcaccac atacgaggac gggggcgtgc 2220
tgaccgctac ccaggacacc agcctccagg acggctgcct catctacaac gtcaagatca 2280
gaggggtgaa cttcacatcc aacggccctg tgatgcagaa gaaaacactc ggctgggagg 2340
ccttcaccga gacgctgtac cccgctgacg gcggcctgga aggcagaaac gacatggccc 2400
tgaagctcgt gggcgggagc catctgatcg caaacatcaa gaccacatat agatccaaga 2460
aacccgctaa gaacctcaag atgcctggcg tctactatgt ggactacaga ctggaaagaa 2520
tcaaggaggc caacaacgag acctacgtcg agcagcacga ggtggcagtg gccagatact 2580
gcgacctccc tagcaaactg gggcacaagc taaattgaaa gctttgcttt atttgtgaaa 2640
tttgtgatgc tattgcttta tttgtaacca ttataagctg caataaacaa gtttaacaac 2700
aacaattgca ttcattttat gtttcaggtt cagggggagg tgtgggaggt tttttaaagt 2760
cgacccacca ttgtctttcc tagcggaatg ggcacctcat cccccgcccg caggggctca 2820
gctgacggcc ctcggagtgc ccagccactg aggcctgagg atggacactg ctcttggccc 2880
ctctgaccgg cttccttggc caccagtgtt ctgcagaccc tccaccatga gcccgggtca 2940
gcgcatttcc tcaggagaag caggcagggt gcaggccatt gcaggccgtc caggggctga 3000
gctgcctggg ggcgaccggg gctccagcct gcacctgcac caggcacagc cccaccacag 3060
gactcatgtc tcaatgccca cagtgagccc aggcagcagg tgtcaccgtc ccctacaggg 3120
agggccagat gcagtcactg cttcaggtcc tgccagcaca gagctgcctg cgtccagctc 3180
cctgaatctc tgctgctgct gctgctgctg ctgctgctgc ctgcggcccg gggctgaagg 3240
cgccgtggcc ctgcctgacg ccccggagcc tcctgcctga acttgggggc tggttggaga 3300
tggccttgga gcagccaagg tgcccctggc agtggcatcc cgaaacgccc tggacgcagg 3360
gcccaagact gggcacagga gtgggaggta catggggctg gggactcccc aggagttatc 3420
tgctccctgc aggcctagag aagtttcagg gaaggtcaga agagctcctg gctgtggtgg 3480
gcagggcagg aaacccctcc acctttacac atgcccaggc agcacctcag gccctttgtg 3540
gggcagggaa gctgaggcag taagcgggca ggcagagctg gaggcctttc aggcccagcc 3600
agcactctgg cctcctgccg ccgcattcca ccccagcccc tcacaccact cgggagaggg 3660
acatcctacg gtcccaaggt caggagggca gggctggggt tgactcaggc ccctcccagc 3720
tgtggccacc tgggtgttgg gagggcagaa gtgcaggcac ctagggcccc ccatgtgccc 3780
accctgggag ctctccttgg aacccattcc tgaaattatt taaaggggtt ggccggacta 3840
gttacgtaga taagtagcat ggcgggttaa tcattaacta caaggaaccc ctagtgatgg 3900
agttggccac tccctctctg cgcgctcgct cgctcactga ggccgggcga ccaaaggtcg 3960
cccgacgccc gggctttgcc cgggcggcct cagtgagcga gcgagcgcgc cagctggcgt 4020
aatagcgaag aggcccgcac cgatcgccct tcccaacagt tgcgcagcct gaatggcgaa 4080
tggcgattcc gttgcaatgg ctggcggtaa tattgttctg gatattacca gcaaggccga 4140
tagtttgagt tcttctactc aggcaagtga tgttattact aatcaaagaa gtattgcgac 4200
aacggttaat ttgcgtgatg gacagactct tttactcggt ggcctcactg attataaaaa 4260
cacttctcag gattctggcg taccgttcct gtctaaaatc cctttaatcg gcctcctgtt 4320
tagctcccgc tctgattcta acgaggaaag cacgttatac gtgctcgtca aagcaaccat 4380
agtacgcgcc ctgtagcggc gcattaagcg cggcgggtgt ggtggttacg cgcagcgtga 4440
ccgctacact tgccagcgcc ctagcgcccg ctcctttcgc tttcttccct tcctttctcg 4500
ccacgttcgc cggctttccc cgtcaagctc taaatcgggg gctcccttta gggttccgat 4560
ttagtgcttt acggcacctc gaccccaaaa aacttgatta gggtgatggt tcacgtagtg 4620
ggccatcgcc ctgatagacg gtttttcgcc ctttgacgtt ggagtccacg ttctttaata 4680
gtggactctt gttccaaact ggaacaacac tcaaccctat ctcggtctat tcttttgatt 4740
tataagggat tttgccgatt tcggcctatt ggttaaaaaa tgagctgatt taacaaaaat 4800
ttaacgcgaa ttttaacaaa atattaacgt ttacaattta aatatttgct tatacaatct 4860
tcctgttttt ggggcttttc tgattatcaa ccggggtaca tatgattgac atgctagttt 4920
tacgattacc gttcatcgat tctcttgttt gctccagact ctcaggcaat gacctgatag 4980
cctttgtaga gacctctcaa aaatagctac cctctccggc atgaatttat cagctagaac 5040
ggttgaatat catattgatg gtgatttgac tgtctccggc ctttctcacc cgtttgaatc 5100
tttacctaca cattactcag gcattgcatt taaaatatat gagggttcta aaaattttta 5160
tccttgcgtt gaaataaagg cttctcccgc aaaagtatta cagggtcata atgtttttgg 5220
tacaaccgat ttagctttat gctctgaggc tttattgctt aattttgcta attctttgcc 5280
ttgcctgtat gatttattgg atgttggaat cgcctgatgc ggtattttct ccttacgcat 5340
ctgtgcggta tttcacaccg catatggtgc actctcagta caatctgctc tgatgccgca 5400
tagttaagcc agccccgaca cccgccaaca cccgctgacg cgccctgacg ggcttgtctg 5460
ctcccggcat ccgcttacag acaagctgtg accgtctccg ggagctgcat gtgtcagagg 5520
ttttcaccgt catcaccgaa acgcgcgaga cgaaagggcc tcgtgatacg cctattttta 5580
taggttaatg tcatgataat aatggtttct tagacgtcag gtggcacttt tcggggaaat 5640
gtgcgcggaa cccctatttg tttatttttc taaatacatt caaatatgta tccgctcatg 5700
agacaataac cctgataaat gcttcaataa tattgaaaaa ggaagagtat gagtattcaa 5760
catttccgtg tcgcccttat tccctttttt gcggcatttt gccttcctgt ttttgctcac 5820
ccagaaacgc tggtgaaagt aaaagatgct gaagatcagt tgggtgcacg agtgggttac 5880
atcgaactgg atctcaacag cggtaagatc cttgagagtt ttcgccccga agaacgtttt 5940
ccaatgatga gcacttttaa agttctgcta tgtggcgcgg tattatcccg tattgacgcc 6000
gggcaagagc aactcggtcg ccgcatacac tattctcaga atgacttggt tgagtactca 6060
ccagtcacag aaaagcatct tacggatggc atgacagtaa gagaattatg cagtgctgcc 6120
ataaccatga gtgataacac tgcggccaac ttacttctga caacgatcgg aggaccgaag 6180
gagctaaccg cttttttgca caacatgggg gatcatgtaa ctcgccttga tcgttgggaa 6240
ccggagctga atgaagccat accaaacgac gagcgtgaca ccacgatgcc tgtagcaatg 6300
gcaacaacgt tgcgcaaact attaactggc gaactactta ctctagcttc ccggcaacaa 6360
ttaatagact ggatggaggc ggataaagtt gcaggaccac ttctgcgctc ggcccttccg 6420
gctggctggt ttattgctga taaatctgga gccggtgagc gtgggtctcg cggtatcatt 6480
gcagcactgg ggccagatgg taagccctcc cgtatcgtag ttatctacac gacggggagt 6540
caggcaacta tggatgaacg aaatagacag atcgctgaga taggtgcctc actgattaag 6600
cattggtaac tgtcagacca agtttactca tatatacttt agattgattt aaaacttcat 6660
ttttaattta aaaggatcta ggtgaagatc ctttttgata atctcatgac caaaatccct 6720
taacgtgagt tttcgttcca ctgagcgtca gaccccgtag aaaagatcaa aggatcttct 6780
tgagatcctt tttttctgcg cgtaatctgc tgcttgcaaa caaaaaaacc accgctacca 6840
gcggtggttt gtttgccgga tcaagagcta ccaactcttt ttccgaaggt aactggcttc 6900
agcagagcgc agataccaaa tactgtcctt ctagtgtagc cgtagttagg ccaccacttc 6960
aagaactctg tagcaccgcc tacatacctc gctctgctaa tcctgttacc agtggctgct 7020
gccagtggcg ataagtcgtg tcttaccggg ttggactcaa gacgatagtt accggataag 7080
gcgcagcggt cgggctgaac ggggggttcg tgcacacagc ccagcttgga gcgaacgacc 7140
tacaccgaac tgagatacct acagcgtgag ctatgagaaa gcgccacgct tcccgaaggg 7200
agaaaggcgg acaggtatcc ggtaagcggc agggtcggaa caggagagcg cacgagggag 7260
cttccagggg gaaacgcctg gtatctttat agtcctgtcg ggtttcgcca cctctgactt 7320
gagcgtcgat ttttgtgatg ctcgtcaggg gggcggagcc tatggaaaaa cgccagcaac 7380
gcggcctttt tacggttcct ggccttttgc tggccttttg ctcacatgtt ctttcctgcg 7440
ttatcccctg attctgtgga taaccgtatt accgcctttg agtgagctga taccgctcgc 7500
cgcagccgaa cgaccgagcg cagcgagtca gtgagcgagg aagcggaaga gcgcccaata 7560
cgcaaaccgc ctctccccgc gcgttggccg attcattaat g 7601
<210>45
<211>1325
<212>ДНК
<213>Homo sapiens
<400>45
agtccagggc tctgtcctgc acctggggaa tggtgaccgg catctctgtc ctctagctct 60
ggaagcaccc cagcccctct agtctgccct cacccctgac cctgaccctc caccctgacc 120
ccgtcctaac ccctgacctt tgtgcccttc cagagagaag ggcagaagtg cccacagccc 180
accccagccc ctcacccagg ccagccggcc agttccaaac cctggtggtt ggtgtcgtgg 240
gcggcctgct gggcagcctg gtgctgctag tctgggtcct ggccgtcatc tgctcccggg 300
ccgcacgagg taacgtcatc ccagcccctc ggcctgccct gccctaaccc tgctggcggc 360
cctcactccc gcctcccctt cctccaccct tccctcaccc caccccacct ccccccatct 420
ccccgccagg ctaagtccct gatgaaggcc cctggactaa gaccccccac ctaggagcac 480
ggctcagggt cggcctggtg accccaagtg tgtttctctg cagggacaat aggagccagg 540
cgcaccggcc agcccctggt gagtctcact cttttcctgc atgatccact gtgccttcct 600
tcctgggtgg gcagaggtgg aaggacaggc tgggaccaca cggcctgcag gactcacatt 660
ctattatagc caggacccca cctccccagc ccccaggcag caacctcaat ccctaaagcc 720
atgatctggg gccccagccc acctgcggtc tccgggggtg cccggcccat gtgtgtgcct 780
gcctgcggtc tccaggggtg cctggcccac gcgtgtgccc gcctgcggtc tctgggggtg 840
cccggcccac atatgtgcct gcctgcggtc tccaggtgtg cccggcccat gcgtgtgccc 900
acctgcgagg gcgtggggtg ggcttggtca tttcttatct tacattggag acaggagagc 960
ttgaaaagtc acattttgga atcctaaatc tgcaagaatg ccagggacat ttcagagggg 1020
gacattgagc cagagaggag gggtggtgtc cccagatcac acagagggca gtggtgggac 1080
agctcagggt aagcagctca tagtgggggg cccaggttcg gtgccggtac tgcagccagg 1140
ctgtggagcc gcgggcctcc ttcctgcggt gggccgtggg gctgactccc tctccctttc 1200
tcctcaaaga aggaggaccc ctcagccgtg cctgtgttct ctgtggacta tggggagctg 1260
gatttccagt ggcgagagaa gaccccggag ccccccgtgc cctgtgtccc tgagcagacg 1320
gagta 1325
<210>46
<211>1072
<212>ДНК
<213>Homo sapiens
<400>46
ccaccattgt ctttcctagc ggaatgggca cctcatcccc cgcccgcagg ggctcagctg 60
acggccctcg gagtgcccag ccactgaggc ctgaggatgg acactgctct tggcccctct 120
gaccggcttc cttggccacc agtgttctgc agaccctcca ccatgagccc gggtcagcgc 180
atttcctcag gagaagcagg cagggtgcag gccattgcag gccgtccagg ggctgagctg 240
cctgggggcg accggggctc cagcctgcac ctgcaccagg cacagcccca ccacaggact 300
catgtctcaa tgcccacagt gagcccaggc agcaggtgtc accgtcccct acagggaggg 360
ccagatgcag tcactgcttc aggtcctgcc agcacagagc tgcctgcgtc cagctccctg 420
aatctctgct gctgctgctg ctgctgctgc tgctgcctgc ggcccggggc tgaaggcgcc 480
gtggccctgc ctgacgcccc ggagcctcct gcctgaactt gggggctggt tggagatggc 540
cttggagcag ccaaggtgcc cctggcagtg gcatcccgaa acgccctgga cgcagggccc 600
aagactgggc acaggagtgg gaggtacatg gggctgggga ctccccagga gttatctgct 660
ccctgcaggc ctagagaagt ttcagggaag gtcagaagag ctcctggctg tggtgggcag 720
ggcaggaaac ccctccacct ttacacatgc ccaggcagca cctcaggccc tttgtggggc 780
agggaagctg aggcagtaag cgggcaggca gagctggagg cctttcaggc ccagccagca 840
ctctggcctc ctgccgccgc attccacccc agcccctcac accactcggg agagggacat 900
cctacggtcc caaggtcagg agggcagggc tggggttgac tcaggcccct cccagctgtg 960
gccacctggg tgttgggagg gcagaagtgc aggcacctag ggccccccat gtgcccaccc 1020
tgggagctct ccttggaacc cattcctgaa attatttaaa ggggttggcc gg 1072
<210>47
<211>7601
<212>ДНК
<213>Искусственная последовательность
<220>
<223>Синтезированная конструкция SNP 3'-гомологичного плеча rAAV PD-1
Exon5_MND-BFP
<400>47
cagctgcgcg ctcgctcgct cactgaggcc gcccgggcaa agcccgggcg tcgggcgacc 60
tttggtcgcc cggcctcagt gagcgagcga gcgcgcagag agggagtggc caactccatc 120
actaggggtt ccttgtagtt aatgattaac ccgccatgct acttatctac gtagaattca 180
gtccagggct ctgtcctgca cctggggaat ggtgaccggc atctctgtcc tctagctctg 240
gaagcacccc agcccctcta gtctgccctc acccctgacc ctgaccctcc accctgaccc 300
cgtcctaacc cctgaccttt gtgcccttcc agagagaagg gcagaagtgc ccacagccca 360
ccccagcccc tcacccaggc cagccggcca gttccaaacc ctggtggttg gtgtcgtggg 420
cggcctgctg ggcagcctgg tgctgctagt ctgggtcctg gccgtcatct gctcccgggc 480
cgcacgaggt aacgtcatcc cagcccctcg gcctgccctg ccctaaccct gctggcggcc 540
ctcactcccg cctccccttc ctccaccctt ccctcacccc accccacctc cccccatctc 600
cccgccaggc taagtccctg atgaaggccc ctggactaag accccccacc taggagcacg 660
gctcagggtc ggcctggtga ccccaagtgt gtttctctgc agggacaata ggagccaggc 720
gcaccggcca gcccctggtg agtctcactc ttttcctgca tgatccactg tgccttcctt 780
cctgggtggg cagaggtgga aggacaggct gggaccacac ggcctgcagg actcacattc 840
tattatagcc aggaccccac ctccccagcc cccaggcagc aacctcaatc cctaaagcca 900
tgatctgggg ccccagccca cctgcggtct ccgggggtgc ccggcccatg tgtgtgcctg 960
cctgcggtct ccaggggtgc ctggcccacg cgtgtgcccg cctgcggtct ctgggggtgc 1020
ccggcccaca tatgtgcctg cctgcggtct ccaggtgtgc ccggcccatg cgtgtgccca 1080
cctgcgaggg cgtggggtgg gcttggtcat ttcttatctt acattggaga caggagagct 1140
tgaaaagtca cattttggaa tcctaaatct gcaagaatgc cagggacatt tcagaggggg 1200
acattgagcc agagaggagg ggtggtgtcc ccagatcaca cagagggcag tggtgggaca 1260
gctcagggta agcagctcat agtggggggc ccaggttcgg tgccggtact gcagccaggc 1320
tgtggagccg cgggcctcct tcctgcggtg ggccgtgggg ctgactccct ctccctttct 1380
cctcaaagaa ggaggacccc tcagccgtgc ctgtgttctc tgtggactat ggggagctgg 1440
atttccagtg gcgagagaag accccggagc cccccgtgcc ctgtgtccct gagcagacgg 1500
agtaatgcat gaacagagaa acaggagaat atgggccaaa caggatatct gtggtaagca 1560
gttcctgccc cggctcaggg ccaagaacag ttggaacagc agaatatggg ccaaacagga 1620
tatctgtggt aagcagttcc tgccccggct cagggccaag aacagatggt ccccagatgc 1680
ggtcccgccc tcagcagttt ctagagaacc atcagatgtt tccagggtgc cccaaggacc 1740
tgaaatgacc ctgtgcctta tttgaactaa ccaatcagtt cgcttctcgc ttctgttcgc 1800
gcgcttctgc tccccgagct ctatataagc agagctcgtt tagtgaaccg tcagatcgcc 1860
tggagacgcc atccacgctg ttttgacttc catagaagga tctcgaggcc accatggcta 1920
gcgagctgat taaggagaac atgcacatga agctgtacat ggagggcacc gtggacaacc 1980
atcacttcaa gtgcacatcc gagggcgaag gcaagcccta cgagggcacc cagaccatga 2040
gaatcaaggt ggtcgagggc ggccctctcc ccttcgcctt cgacatcctg gctactagct 2100
tcctctacgg cagcaagacc ttcatcaacc acacccaggg catccccgac ttcttcaagc 2160
agtccttccc tgagggcttc acatgggaga gagtcaccac atacgaggac gggggcgtgc 2220
tgaccgctac ccaggacacc agcctccagg acggctgcct catctacaac gtcaagatca 2280
gaggggtgaa cttcacatcc aacggccctg tgatgcagaa gaaaacactc ggctgggagg 2340
ccttcaccga gacgctgtac cccgctgacg gcggcctgga aggcagaaac gacatggccc 2400
tgaagctcgt gggcgggagc catctgatcg caaacatcaa gaccacatat agatccaaga 2460
aacccgctaa gaacctcaag atgcctggcg tctactatgt ggactacaga ctggaaagaa 2520
tcaaggaggc caacaacgag acctacgtcg agcagcacga ggtggcagtg gccagatact 2580
gcgacctccc tagcaaactg gggcacaagc taaattgaaa gctttgcttt atttgtgaaa 2640
tttgtgatgc tattgcttta tttgtaacca ttataagctg caataaacaa gtttaacaac 2700
aacaattgca ttcattttat gtttcaggtt cagggggagg tgtgggaggt tttttaaagt 2760
cgacccacca ttgtctttcc tagcggaatg ggcacctcat cccccgcccg caggggctca 2820
gccgacggcc ctcggagtgc ccagccactg aggcctgagg atggacactg ctcttggccc 2880
ctctgaccgg cttccttggc caccagtgtt ctgcagaccc tccaccatga gcccgggtca 2940
gcgcatttcc tcaggagaag caggcagggt gcaggccatt gcaggccgtc caggggctga 3000
gctgcctggg ggcgaccggg gctccagcct gcacctgcac caggcacagc cccaccacag 3060
gactcatgtc tcaatgccca cagtgagccc aggcagcagg tgtcaccgtc ccctacaggg 3120
agggccagat gcagtcactg cttcaggtcc tgccagcaca gagctgcctg cgtccagctc 3180
cctgaatctc tgctgctgct gctgctgctg ctgctgctgc ctgcggcccg gggctgaagg 3240
cgccgtggcc ctgcctgacg ccccggagcc tcctgcctga acttgggggc tggttggaga 3300
tggccttgga gcagccaagg tgcccctggc agtggcatcc cgaaacgccc tggacgcagg 3360
gcccaagact gggcacagga gtgggaggta catggggctg gggactcccc aggagttatc 3420
tgctccctgc aggcctagag aagtttcagg gaaggtcaga agagctcctg gctgtggtgg 3480
gcagggcagg aaacccctcc acctttacac atgcccaggc agcacctcag gccctttgtg 3540
gggcagggaa gctgaggcag taagcgggca ggcagagctg gaggcctttc aggcccagcc 3600
agcactctgg cctcctgccg ccgcattcca ccccagcccc tcacaccact cgggagaggg 3660
acatcctacg gtcccaaggt caggagggca gggctggggt tgactcaggc ccctcccagc 3720
tgtggccacc tgggtgttgg gagggcagaa gtgcaggcac ctagggcccc ccatgtgccc 3780
accctgggag ctctccttgg aacccattcc tgaaattatt taaaggggtt ggccggacta 3840
gttacgtaga taagtagcat ggcgggttaa tcattaacta caaggaaccc ctagtgatgg 3900
agttggccac tccctctctg cgcgctcgct cgctcactga ggccgggcga ccaaaggtcg 3960
cccgacgccc gggctttgcc cgggcggcct cagtgagcga gcgagcgcgc cagctggcgt 4020
aatagcgaag aggcccgcac cgatcgccct tcccaacagt tgcgcagcct gaatggcgaa 4080
tggcgattcc gttgcaatgg ctggcggtaa tattgttctg gatattacca gcaaggccga 4140
tagtttgagt tcttctactc aggcaagtga tgttattact aatcaaagaa gtattgcgac 4200
aacggttaat ttgcgtgatg gacagactct tttactcggt ggcctcactg attataaaaa 4260
cacttctcag gattctggcg taccgttcct gtctaaaatc cctttaatcg gcctcctgtt 4320
tagctcccgc tctgattcta acgaggaaag cacgttatac gtgctcgtca aagcaaccat 4380
agtacgcgcc ctgtagcggc gcattaagcg cggcgggtgt ggtggttacg cgcagcgtga 4440
ccgctacact tgccagcgcc ctagcgcccg ctcctttcgc tttcttccct tcctttctcg 4500
ccacgttcgc cggctttccc cgtcaagctc taaatcgggg gctcccttta gggttccgat 4560
ttagtgcttt acggcacctc gaccccaaaa aacttgatta gggtgatggt tcacgtagtg 4620
ggccatcgcc ctgatagacg gtttttcgcc ctttgacgtt ggagtccacg ttctttaata 4680
gtggactctt gttccaaact ggaacaacac tcaaccctat ctcggtctat tcttttgatt 4740
tataagggat tttgccgatt tcggcctatt ggttaaaaaa tgagctgatt taacaaaaat 4800
ttaacgcgaa ttttaacaaa atattaacgt ttacaattta aatatttgct tatacaatct 4860
tcctgttttt ggggcttttc tgattatcaa ccggggtaca tatgattgac atgctagttt 4920
tacgattacc gttcatcgat tctcttgttt gctccagact ctcaggcaat gacctgatag 4980
cctttgtaga gacctctcaa aaatagctac cctctccggc atgaatttat cagctagaac 5040
ggttgaatat catattgatg gtgatttgac tgtctccggc ctttctcacc cgtttgaatc 5100
tttacctaca cattactcag gcattgcatt taaaatatat gagggttcta aaaattttta 5160
tccttgcgtt gaaataaagg cttctcccgc aaaagtatta cagggtcata atgtttttgg 5220
tacaaccgat ttagctttat gctctgaggc tttattgctt aattttgcta attctttgcc 5280
ttgcctgtat gatttattgg atgttggaat cgcctgatgc ggtattttct ccttacgcat 5340
ctgtgcggta tttcacaccg catatggtgc actctcagta caatctgctc tgatgccgca 5400
tagttaagcc agccccgaca cccgccaaca cccgctgacg cgccctgacg ggcttgtctg 5460
ctcccggcat ccgcttacag acaagctgtg accgtctccg ggagctgcat gtgtcagagg 5520
ttttcaccgt catcaccgaa acgcgcgaga cgaaagggcc tcgtgatacg cctattttta 5580
taggttaatg tcatgataat aatggtttct tagacgtcag gtggcacttt tcggggaaat 5640
gtgcgcggaa cccctatttg tttatttttc taaatacatt caaatatgta tccgctcatg 5700
agacaataac cctgataaat gcttcaataa tattgaaaaa ggaagagtat gagtattcaa 5760
catttccgtg tcgcccttat tccctttttt gcggcatttt gccttcctgt ttttgctcac 5820
ccagaaacgc tggtgaaagt aaaagatgct gaagatcagt tgggtgcacg agtgggttac 5880
atcgaactgg atctcaacag cggtaagatc cttgagagtt ttcgccccga agaacgtttt 5940
ccaatgatga gcacttttaa agttctgcta tgtggcgcgg tattatcccg tattgacgcc 6000
gggcaagagc aactcggtcg ccgcatacac tattctcaga atgacttggt tgagtactca 6060
ccagtcacag aaaagcatct tacggatggc atgacagtaa gagaattatg cagtgctgcc 6120
ataaccatga gtgataacac tgcggccaac ttacttctga caacgatcgg aggaccgaag 6180
gagctaaccg cttttttgca caacatgggg gatcatgtaa ctcgccttga tcgttgggaa 6240
ccggagctga atgaagccat accaaacgac gagcgtgaca ccacgatgcc tgtagcaatg 6300
gcaacaacgt tgcgcaaact attaactggc gaactactta ctctagcttc ccggcaacaa 6360
ttaatagact ggatggaggc ggataaagtt gcaggaccac ttctgcgctc ggcccttccg 6420
gctggctggt ttattgctga taaatctgga gccggtgagc gtgggtctcg cggtatcatt 6480
gcagcactgg ggccagatgg taagccctcc cgtatcgtag ttatctacac gacggggagt 6540
caggcaacta tggatgaacg aaatagacag atcgctgaga taggtgcctc actgattaag 6600
cattggtaac tgtcagacca agtttactca tatatacttt agattgattt aaaacttcat 6660
ttttaattta aaaggatcta ggtgaagatc ctttttgata atctcatgac caaaatccct 6720
taacgtgagt tttcgttcca ctgagcgtca gaccccgtag aaaagatcaa aggatcttct 6780
tgagatcctt tttttctgcg cgtaatctgc tgcttgcaaa caaaaaaacc accgctacca 6840
gcggtggttt gtttgccgga tcaagagcta ccaactcttt ttccgaaggt aactggcttc 6900
agcagagcgc agataccaaa tactgtcctt ctagtgtagc cgtagttagg ccaccacttc 6960
aagaactctg tagcaccgcc tacatacctc gctctgctaa tcctgttacc agtggctgct 7020
gccagtggcg ataagtcgtg tcttaccggg ttggactcaa gacgatagtt accggataag 7080
gcgcagcggt cgggctgaac ggggggttcg tgcacacagc ccagcttgga gcgaacgacc 7140
tacaccgaac tgagatacct acagcgtgag ctatgagaaa gcgccacgct tcccgaaggg 7200
agaaaggcgg acaggtatcc ggtaagcggc agggtcggaa caggagagcg cacgagggag 7260
cttccagggg gaaacgcctg gtatctttat agtcctgtcg ggtttcgcca cctctgactt 7320
gagcgtcgat ttttgtgatg ctcgtcaggg gggcggagcc tatggaaaaa cgccagcaac 7380
gcggcctttt tacggttcct ggccttttgc tggccttttg ctcacatgtt ctttcctgcg 7440
ttatcccctg attctgtgga taaccgtatt accgcctttg agtgagctga taccgctcgc 7500
cgcagccgaa cgaccgagcg cagcgagtca gtgagcgagg aagcggaaga gcgcccaata 7560
cgcaaaccgc ctctccccgc gcgttggccg attcattaat g 7601
<210>48
<211>7601
<212>ДНК
<213>Искусственная последовательность
<220>
<223>Синтезированная конструкция SNP rAAV PD-1 Exon5_MND-BFP 5'-
гомологичное плечо
<400>48
cagctgcgcg ctcgctcgct cactgaggcc gcccgggcaa agcccgggcg tcgggcgacc 60
tttggtcgcc cggcctcagt gagcgagcga gcgcgcagag agggagtggc caactccatc 120
actaggggtt ccttgtagtt aatgattaac ccgccatgct acttatctac gtagaattca 180
gtccagggct ctgtcctgca cctggggaat ggtgaccggc atctctgtcc tctagctctg 240
gaagcacccc agcccctcta gtctgccctc acccctgacc ctgaccctcc accctgaccc 300
cgtcctaacc cctgaccttt gtgcccttcc agagagaagg gcagaagtgc ccacagccca 360
ccccagcccc tcacccaggc cagccggcca gttccaaacc ctggtggttg gtgtcgtggg 420
cggcctgctg ggcagcctgg tgctgctagt ctgggtcctg gccgtcatct gctcccgggc 480
cgcacgaggt aacgtcatcc cagcccctcg gcctgccctg ccctaaccct gctggcggcc 540
ctcactcccg cctccccttc ctccaccctt ccctcacccc accccacctc cccccatctc 600
cccgccaggc taagtccctg atgaaggccc ctggactaag accccccacc taggagcacg 660
gctcagggtc ggcctggtga ccccaagtgt gtttctctgc agggacaata ggagccaggc 720
gcaccggcca gcccctggtg agtctcactc ttttcctgca tgatccactg tgccttcctt 780
cctgggtggg cagaggtgga aggacaggct gggaccacac ggcctgcagg actcacattc 840
tattatagcc aggaccccac ctccccagcc cccaggcagc aacctcaatc cctaaagcca 900
tgatctgggg ccccagccca cctgcggtct ccgggggtgc ccggcccatg tgtgtgcctg 960
cctgcggtct ccaggggtgc ctggcccacg cgtgtgcccg cctgcggtct ctgggggtgc 1020
ccggcccaca tatgtgcctg cctgcggtct ccaggtgtgc ccggcccatg cgtgtgccca 1080
cctgcgaggg cgtggggtgg gcttggtcat ttcttatctt acattggaga caggagagct 1140
tgaaaagtca cattttggaa tcctaaatct gcaagaatgc cagggacatt tcagaggggg 1200
acattgagcc agagaggagg ggtggtgtcc ccagatcaca cagagggcag tggtgggaca 1260
gctcagggta agcagctcgt agtggggggc ccaggttcgg tgccggtact gcagccaggc 1320
tgtggagccg cgggcctcct tcctgcggtg ggccgtgggg ctgactccct ctccctttct 1380
cctcaaagaa ggaggacccc tcagccgtgc ctgtgttctc tgtggactat ggggagctgg 1440
atttccagtg gcgagagaag accccggagc cccccgtgcc ctgtgtccct gagcagacgg 1500
agtaatgcat gaacagagaa acaggagaat atgggccaaa caggatatct gtggtaagca 1560
gttcctgccc cggctcaggg ccaagaacag ttggaacagc agaatatggg ccaaacagga 1620
tatctgtggt aagcagttcc tgccccggct cagggccaag aacagatggt ccccagatgc 1680
ggtcccgccc tcagcagttt ctagagaacc atcagatgtt tccagggtgc cccaaggacc 1740
tgaaatgacc ctgtgcctta tttgaactaa ccaatcagtt cgcttctcgc ttctgttcgc 1800
gcgcttctgc tccccgagct ctatataagc agagctcgtt tagtgaaccg tcagatcgcc 1860
tggagacgcc atccacgctg ttttgacttc catagaagga tctcgaggcc accatggcta 1920
gcgagctgat taaggagaac atgcacatga agctgtacat ggagggcacc gtggacaacc 1980
atcacttcaa gtgcacatcc gagggcgaag gcaagcccta cgagggcacc cagaccatga 2040
gaatcaaggt ggtcgagggc ggccctctcc ccttcgcctt cgacatcctg gctactagct 2100
tcctctacgg cagcaagacc ttcatcaacc acacccaggg catccccgac ttcttcaagc 2160
agtccttccc tgagggcttc acatgggaga gagtcaccac atacgaggac gggggcgtgc 2220
tgaccgctac ccaggacacc agcctccagg acggctgcct catctacaac gtcaagatca 2280
gaggggtgaa cttcacatcc aacggccctg tgatgcagaa gaaaacactc ggctgggagg 2340
ccttcaccga gacgctgtac cccgctgacg gcggcctgga aggcagaaac gacatggccc 2400
tgaagctcgt gggcgggagc catctgatcg caaacatcaa gaccacatat agatccaaga 2460
aacccgctaa gaacctcaag atgcctggcg tctactatgt ggactacaga ctggaaagaa 2520
tcaaggaggc caacaacgag acctacgtcg agcagcacga ggtggcagtg gccagatact 2580
gcgacctccc tagcaaactg gggcacaagc taaattgaaa gctttgcttt atttgtgaaa 2640
tttgtgatgc tattgcttta tttgtaacca ttataagctg caataaacaa gtttaacaac 2700
aacaattgca ttcattttat gtttcaggtt cagggggagg tgtgggaggt tttttaaagt 2760
cgacccacca ttgtctttcc tagcggaatg ggcacctcat cccccgcccg caggggctca 2820
gctgacggcc ctcggagtgc ccagccactg aggcctgagg atggacactg ctcttggccc 2880
ctctgaccgg cttccttggc caccagtgtt ctgcagaccc tccaccatga gcccgggtca 2940
gcgcatttcc tcaggagaag caggcagggt gcaggccatt gcaggccgtc caggggctga 3000
gctgcctggg ggcgaccggg gctccagcct gcacctgcac caggcacagc cccaccacag 3060
gactcatgtc tcaatgccca cagtgagccc aggcagcagg tgtcaccgtc ccctacaggg 3120
agggccagat gcagtcactg cttcaggtcc tgccagcaca gagctgcctg cgtccagctc 3180
cctgaatctc tgctgctgct gctgctgctg ctgctgctgc ctgcggcccg gggctgaagg 3240
cgccgtggcc ctgcctgacg ccccggagcc tcctgcctga acttgggggc tggttggaga 3300
tggccttgga gcagccaagg tgcccctggc agtggcatcc cgaaacgccc tggacgcagg 3360
gcccaagact gggcacagga gtgggaggta catggggctg gggactcccc aggagttatc 3420
tgctccctgc aggcctagag aagtttcagg gaaggtcaga agagctcctg gctgtggtgg 3480
gcagggcagg aaacccctcc acctttacac atgcccaggc agcacctcag gccctttgtg 3540
gggcagggaa gctgaggcag taagcgggca ggcagagctg gaggcctttc aggcccagcc 3600
agcactctgg cctcctgccg ccgcattcca ccccagcccc tcacaccact cgggagaggg 3660
acatcctacg gtcccaaggt caggagggca gggctggggt tgactcaggc ccctcccagc 3720
tgtggccacc tgggtgttgg gagggcagaa gtgcaggcac ctagggcccc ccatgtgccc 3780
accctgggag ctctccttgg aacccattcc tgaaattatt taaaggggtt ggccggacta 3840
gttacgtaga taagtagcat ggcgggttaa tcattaacta caaggaaccc ctagtgatgg 3900
agttggccac tccctctctg cgcgctcgct cgctcactga ggccgggcga ccaaaggtcg 3960
cccgacgccc gggctttgcc cgggcggcct cagtgagcga gcgagcgcgc cagctggcgt 4020
aatagcgaag aggcccgcac cgatcgccct tcccaacagt tgcgcagcct gaatggcgaa 4080
tggcgattcc gttgcaatgg ctggcggtaa tattgttctg gatattacca gcaaggccga 4140
tagtttgagt tcttctactc aggcaagtga tgttattact aatcaaagaa gtattgcgac 4200
aacggttaat ttgcgtgatg gacagactct tttactcggt ggcctcactg attataaaaa 4260
cacttctcag gattctggcg taccgttcct gtctaaaatc cctttaatcg gcctcctgtt 4320
tagctcccgc tctgattcta acgaggaaag cacgttatac gtgctcgtca aagcaaccat 4380
agtacgcgcc ctgtagcggc gcattaagcg cggcgggtgt ggtggttacg cgcagcgtga 4440
ccgctacact tgccagcgcc ctagcgcccg ctcctttcgc tttcttccct tcctttctcg 4500
ccacgttcgc cggctttccc cgtcaagctc taaatcgggg gctcccttta gggttccgat 4560
ttagtgcttt acggcacctc gaccccaaaa aacttgatta gggtgatggt tcacgtagtg 4620
ggccatcgcc ctgatagacg gtttttcgcc ctttgacgtt ggagtccacg ttctttaata 4680
gtggactctt gttccaaact ggaacaacac tcaaccctat ctcggtctat tcttttgatt 4740
tataagggat tttgccgatt tcggcctatt ggttaaaaaa tgagctgatt taacaaaaat 4800
ttaacgcgaa ttttaacaaa atattaacgt ttacaattta aatatttgct tatacaatct 4860
tcctgttttt ggggcttttc tgattatcaa ccggggtaca tatgattgac atgctagttt 4920
tacgattacc gttcatcgat tctcttgttt gctccagact ctcaggcaat gacctgatag 4980
cctttgtaga gacctctcaa aaatagctac cctctccggc atgaatttat cagctagaac 5040
ggttgaatat catattgatg gtgatttgac tgtctccggc ctttctcacc cgtttgaatc 5100
tttacctaca cattactcag gcattgcatt taaaatatat gagggttcta aaaattttta 5160
tccttgcgtt gaaataaagg cttctcccgc aaaagtatta cagggtcata atgtttttgg 5220
tacaaccgat ttagctttat gctctgaggc tttattgctt aattttgcta attctttgcc 5280
ttgcctgtat gatttattgg atgttggaat cgcctgatgc ggtattttct ccttacgcat 5340
ctgtgcggta tttcacaccg catatggtgc actctcagta caatctgctc tgatgccgca 5400
tagttaagcc agccccgaca cccgccaaca cccgctgacg cgccctgacg ggcttgtctg 5460
ctcccggcat ccgcttacag acaagctgtg accgtctccg ggagctgcat gtgtcagagg 5520
ttttcaccgt catcaccgaa acgcgcgaga cgaaagggcc tcgtgatacg cctattttta 5580
taggttaatg tcatgataat aatggtttct tagacgtcag gtggcacttt tcggggaaat 5640
gtgcgcggaa cccctatttg tttatttttc taaatacatt caaatatgta tccgctcatg 5700
agacaataac cctgataaat gcttcaataa tattgaaaaa ggaagagtat gagtattcaa 5760
catttccgtg tcgcccttat tccctttttt gcggcatttt gccttcctgt ttttgctcac 5820
ccagaaacgc tggtgaaagt aaaagatgct gaagatcagt tgggtgcacg agtgggttac 5880
atcgaactgg atctcaacag cggtaagatc cttgagagtt ttcgccccga agaacgtttt 5940
ccaatgatga gcacttttaa agttctgcta tgtggcgcgg tattatcccg tattgacgcc 6000
gggcaagagc aactcggtcg ccgcatacac tattctcaga atgacttggt tgagtactca 6060
ccagtcacag aaaagcatct tacggatggc atgacagtaa gagaattatg cagtgctgcc 6120
ataaccatga gtgataacac tgcggccaac ttacttctga caacgatcgg aggaccgaag 6180
gagctaaccg cttttttgca caacatgggg gatcatgtaa ctcgccttga tcgttgggaa 6240
ccggagctga atgaagccat accaaacgac gagcgtgaca ccacgatgcc tgtagcaatg 6300
gcaacaacgt tgcgcaaact attaactggc gaactactta ctctagcttc ccggcaacaa 6360
ttaatagact ggatggaggc ggataaagtt gcaggaccac ttctgcgctc ggcccttccg 6420
gctggctggt ttattgctga taaatctgga gccggtgagc gtgggtctcg cggtatcatt 6480
gcagcactgg ggccagatgg taagccctcc cgtatcgtag ttatctacac gacggggagt 6540
caggcaacta tggatgaacg aaatagacag atcgctgaga taggtgcctc actgattaag 6600
cattggtaac tgtcagacca agtttactca tatatacttt agattgattt aaaacttcat 6660
ttttaattta aaaggatcta ggtgaagatc ctttttgata atctcatgac caaaatccct 6720
taacgtgagt tttcgttcca ctgagcgtca gaccccgtag aaaagatcaa aggatcttct 6780
tgagatcctt tttttctgcg cgtaatctgc tgcttgcaaa caaaaaaacc accgctacca 6840
gcggtggttt gtttgccgga tcaagagcta ccaactcttt ttccgaaggt aactggcttc 6900
agcagagcgc agataccaaa tactgtcctt ctagtgtagc cgtagttagg ccaccacttc 6960
aagaactctg tagcaccgcc tacatacctc gctctgctaa tcctgttacc agtggctgct 7020
gccagtggcg ataagtcgtg tcttaccggg ttggactcaa gacgatagtt accggataag 7080
gcgcagcggt cgggctgaac ggggggttcg tgcacacagc ccagcttgga gcgaacgacc 7140
tacaccgaac tgagatacct acagcgtgag ctatgagaaa gcgccacgct tcccgaaggg 7200
agaaaggcgg acaggtatcc ggtaagcggc agggtcggaa caggagagcg cacgagggag 7260
cttccagggg gaaacgcctg gtatctttat agtcctgtcg ggtttcgcca cctctgactt 7320
gagcgtcgat ttttgtgatg ctcgtcaggg gggcggagcc tatggaaaaa cgccagcaac 7380
gcggcctttt tacggttcct ggccttttgc tggccttttg ctcacatgtt ctttcctgcg 7440
ttatcccctg attctgtgga taaccgtatt accgcctttg agtgagctga taccgctcgc 7500
cgcagccgaa cgaccgagcg cagcgagtca gtgagcgagg aagcggaaga gcgcccaata 7560
cgcaaaccgc ctctccccgc gcgttggccg attcattaat g 7601
<210>49
<211>1325
<212>ДНК
<213>Homo sapiens
<400>49
agtccagggc tctgtcctgc acctggggaa tggtgaccgg catctctgtc ctctagctct 60
ggaagcaccc cagcccctct agtctgccct cacccctgac cctgaccctc caccctgacc 120
ccgtcctaac ccctgacctt tgtgcccttc cagagagaag ggcagaagtg cccacagccc 180
accccagccc ctcacccagg ccagccggcc agttccaaac cctggtggtt ggtgtcgtgg 240
gcggcctgct gggcagcctg gtgctgctag tctgggtcct ggccgtcatc tgctcccggg 300
ccgcacgagg taacgtcatc ccagcccctc ggcctgccct gccctaaccc tgctggcggc 360
cctcactccc gcctcccctt cctccaccct tccctcaccc caccccacct ccccccatct 420
ccccgccagg ctaagtccct gatgaaggcc cctggactaa gaccccccac ctaggagcac 480
ggctcagggt cggcctggtg accccaagtg tgtttctctg cagggacaat aggagccagg 540
cgcaccggcc agcccctggt gagtctcact cttttcctgc atgatccact gtgccttcct 600
tcctgggtgg gcagaggtgg aaggacaggc tgggaccaca cggcctgcag gactcacatt 660
ctattatagc caggacccca cctccccagc ccccaggcag caacctcaat ccctaaagcc 720
atgatctggg gccccagccc acctgcggtc tccgggggtg cccggcccat gtgtgtgcct 780
gcctgcggtc tccaggggtg cctggcccac gcgtgtgccc gcctgcggtc tctgggggtg 840
cccggcccac atatgtgcct gcctgcggtc tccaggtgtg cccggcccat gcgtgtgccc 900
acctgcgagg gcgtggggtg ggcttggtca tttcttatct tacattggag acaggagagc 960
ttgaaaagtc acattttgga atcctaaatc tgcaagaatg ccagggacat ttcagagggg 1020
gacattgagc cagagaggag gggtggtgtc cccagatcac acagagggca gtggtgggac 1080
agctcagggt aagcagctcg tagtgggggg cccaggttcg gtgccggtac tgcagccagg 1140
ctgtggagcc gcgggcctcc ttcctgcggt gggccgtggg gctgactccc tctccctttc 1200
tcctcaaaga aggaggaccc ctcagccgtg cctgtgttct ctgtggacta tggggagctg 1260
gatttccagt ggcgagagaa gaccccggag ccccccgtgc cctgtgtccc tgagcagacg 1320
gagta 1325
<210>50
<211>1072
<212>ДНК
<213>Homo sapiens
<400>50
ccaccattgt ctttcctagc ggaatgggca cctcatcccc cgcccgcagg ggctcagccg 60
acggccctcg gagtgcccag ccactgaggc ctgaggatgg acactgctct tggcccctct 120
gaccggcttc cttggccacc agtgttctgc agaccctcca ccatgagccc gggtcagcgc 180
atttcctcag gagaagcagg cagggtgcag gccattgcag gccgtccagg ggctgagctg 240
cctgggggcg accggggctc cagcctgcac ctgcaccagg cacagcccca ccacaggact 300
catgtctcaa tgcccacagt gagcccaggc agcaggtgtc accgtcccct acagggaggg 360
ccagatgcag tcactgcttc aggtcctgcc agcacagagc tgcctgcgtc cagctccctg 420
aatctctgct gctgctgctg ctgctgctgc tgctgcctgc ggcccggggc tgaaggcgcc 480
gtggccctgc ctgacgcccc ggagcctcct gcctgaactt gggggctggt tggagatggc 540
cttggagcag ccaaggtgcc cctggcagtg gcatcccgaa acgccctgga cgcagggccc 600
aagactgggc acaggagtgg gaggtacatg gggctgggga ctccccagga gttatctgct 660
ccctgcaggc ctagagaagt ttcagggaag gtcagaagag ctcctggctg tggtgggcag 720
ggcaggaaac ccctccacct ttacacatgc ccaggcagca cctcaggccc tttgtggggc 780
agggaagctg aggcagtaag cgggcaggca gagctggagg cctttcaggc ccagccagca 840
ctctggcctc ctgccgccgc attccacccc agcccctcac accactcggg agagggacat 900
cctacggtcc caaggtcagg agggcagggc tggggttgac tcaggcccct cccagctgtg 960
gccacctggg tgttgggagg gcagaagtgc aggcacctag ggccccccat gtgcccaccc 1020
tgggagctct ccttggaacc cattcctgaa attatttaaa ggggttggcc gg 1072
<210>51
<211>7880
<212>ДНК
<213>Искусственная последовательность
<220>
<223>Синтезированная конструкция рецептора-переключателя rAAV PD-1
Exon5_MND-PD-1-CD28
<400>51
cagctgcgcg ctcgctcgct cactgaggcc gcccgggcaa agcccgggcg tcgggcgacc 60
tttggtcgcc cggcctcagt gagcgagcga gcgcgcagag agggagtggc caactccatc 120
actaggggtt ccttgtagtt aatgattaac ccgccatgct acttatctac gtagaattca 180
gtccagggct ctgtcctgca cctggggaat ggtgaccggc atctctgtcc tctagctctg 240
gaagcacccc agcccctcta gtctgccctc acccctgacc ctgaccctcc accctgaccc 300
cgtcctaacc cctgaccttt gtgcccttcc agagagaagg gcagaagtgc ccacagccca 360
ccccagcccc tcacccaggc cagccggcca gttccaaacc ctggtggttg gtgtcgtggg 420
cggcctgctg ggcagcctgg tgctgctagt ctgggtcctg gccgtcatct gctcccgggc 480
cgcacgaggt aacgtcatcc cagcccctcg gcctgccctg ccctaaccct gctggcggcc 540
ctcactcccg cctccccttc ctccaccctt ccctcacccc accccacctc cccccatctc 600
cccgccaggc taagtccctg atgaaggccc ctggactaag accccccacc taggagcacg 660
gctcagggtc ggcctggtga ccccaagtgt gtttctctgc agggacaata ggagccaggc 720
gcaccggcca gcccctggtg agtctcactc ttttcctgca tgatccactg tgccttcctt 780
cctgggtggg cagaggtgga aggacaggct gggaccacac ggcctgcagg actcacattc 840
tattatagcc aggaccccac ctccccagcc cccaggcagc aacctcaatc cctaaagcca 900
tgatctgggg ccccagccca cctgcggtct ccgggggtgc ccggcccatg tgtgtgcctg 960
cctgcggtct ccaggggtgc ctggcccacg cgtgtgcccg cctgcggtct ctgggggtgc 1020
ccggcccaca tatgtgcctg cctgcggtct ccaggtgtgc ccggcccatg cgtgtgccca 1080
cctgcgaggg cgtggggtgg gcttggtcat ttcttatctt acattggaga caggagagct 1140
tgaaaagtca cattttggaa tcctaaatct gcaagaatgc cagggacatt tcagaggggg 1200
acattgagcc agagaggagg ggtggtgtcc ccagatcaca cagagggcag tggtgggaca 1260
gctcagggta agcagctcat agtggggggc ccaggttcgg tgccggtact gcagccaggc 1320
tgtggagccg cgggcctcct tcctgcggtg ggccgtgggg ctgactccct ctccctttct 1380
cctcaaagaa ggaggacccc tcagccgtgc ctgtgttctc tgtggactat ggggagctgg 1440
atttccagtg gcgagagaag accccggagc cccccgtgcc ctgtgtccct gagcagacgg 1500
agtaatgcat gaacagagaa acaggagaat atgggccaaa caggatatct gtggtaagca 1560
gttcctgccc cggctcaggg ccaagaacag ttggaacagc agaatatggg ccaaacagga 1620
tatctgtggt aagcagttcc tgccccggct cagggccaag aacagatggt ccccagatgc 1680
ggtcccgccc tcagcagttt ctagagaacc atcagatgtt tccagggtgc cccaaggacc 1740
tgaaatgacc ctgtgcctta tttgaactaa ccaatcagtt cgcttctcgc ttctgttcgc 1800
gcgcttctgc tccccgagct ctatataagc agagctcgtt tagtgaaccg tcagatcgcc 1860
tggagacgcc atccacgctg ttttgacttc catagaagga tctcgaggcc accatgcaga 1920
tcccgcaagc gccctggcca gtcgtctggg cggtgctaca actgggctgg cggccaggat 1980
ggttcttaga ctccccagac aggccctgga acccccccac cttctcccca gccctgctcg 2040
tggtgaccga aggggacaac gccaccttca cctgcagctt ctccaacaca tcggagagct 2100
tcgtgctaaa ctggtaccgc atgagcccca gcaaccagac ggacaagctg gccgccttcc 2160
ccgaggaccg cagccagccc ggccaggact gccgcttccg tgtcacacaa ctgcccaacg 2220
ggcgtgactt ccacatgagc gtggtcaggg cccggcgcaa tgacagcggc acctacctct 2280
gtggggccat ctccctggcc cccaaggcgc agatcaaaga gagcctgcgg gcagagctca 2340
gggtgacaga gagaagggca gaagtgccca cagcccaccc cagcccctca cccaggccag 2400
ccggccagtt ccaaaccctg gtggttggtg tcgtgggcgg cctgctgggc agcctggtgc 2460
tgctagtctg ggtcctggcc gtcatcagga gtaagaggag caggctcctg cacagtgact 2520
acatgaacat gactccccgc cgccccgggc ccacccgcaa gcattaccag ccctatgccc 2580
caccacgcga cttcgcagcc tatcgctccg gtgagggcag aggaagtctt ctaacatgcg 2640
gtgacgtgga ggagaatccg ggccctgtga gcaagggcga ggaggataac tccgccatca 2700
tcaaggagtt cctgcgcttc aaggtgcaca tggagggctc cgtgaacggc cacgagttcg 2760
agatcgaggg cgagggcgag ggccgcccct acgagggcac ccagaccgcc aagctgaagg 2820
tgaccaaggg tggccccctg cccttcgcct gggacatcct gtcccctcag ttcatgtacg 2880
gctccaaggc ctacgtgaag caccccgccg acatccccga ctacttgaag ctgtccttcc 2940
ccgagggctt caagtgggag cgcgtgatga acttcgagga cggcggcgtg gtgaccgtga 3000
cccaggactc ctctctgcag gacggcgagt tcatctacaa ggtgaagctg cgcggcacca 3060
acttcccctc cgacggcccc gtaatgcaga agaagaccat gggctgggag gcctcctccg 3120
agcggatgta ccccgaggac ggcgccctga agggcgagat caagcagagg ctgaagctga 3180
aggacggcgg ccactacgac gctgaggtca agaccaccta caaggccaag aagcccgtgc 3240
agctgcccgg cgcctacaac gtcaacatca agttggacat cacctcccac aacgaggact 3300
acaccatcgt ggaacagtac gaacgcgccg agggccgcca ctccaccggc ggcatggacg 3360
agctgtacaa gtgatgaaag ctttgcttta tttgtgaaat ttgtgatgct attgctttat 3420
ttgtaaccat tataagctgc aataaacaag tttaacaaca acaattgcat tcattttatg 3480
tttcaggttc agggggaggt gtgggaggtt ttttaaagtc gacgtggccc tgcctgacgc 3540
cccggagcct cctgcctgaa cttgggggct ggttggagat ggccttggag cagccaaggt 3600
gcccctggca gtggcatccc gaaacgccct ggacgcaggg cccaagactg ggcacaggag 3660
tgggaggtac atggggctgg ggactcccca ggagttatct gctccctgca ggcctagaga 3720
agtttcaggg aaggtcagaa gagctcctgg ctgtggtggg cagggcagga aacccctcca 3780
cctttacaca tgcccaggca gcacctcagg ccctttgtgg ggcagggaag ctgaggcagt 3840
aagcgggcag gcagagctgg aggcctttca ggcccagcca gcactctggc ctcctgccgc 3900
cgcattccac cccagcccct cacaccactc gggagaggga catcctacgg tcccaaggtc 3960
aggagggcag ggctggggtt gactcaggcc cctcccagct gtggccacct gggtgttggg 4020
agggcagaag tgcaggcacc tagggccccc catgtgccca ccctgggagc tctccttgga 4080
acccattcct gaaattattt aaaggggttg gccggactag ttacgtagat aagtagcatg 4140
gcgggttaat cattaactac aaggaacccc tagtgatgga gttggccact ccctctctgc 4200
gcgctcgctc gctcactgag gccgggcgac caaaggtcgc ccgacgcccg ggctttgccc 4260
gggcggcctc agtgagcgag cgagcgcgcc agctggcgta atagcgaaga ggcccgcacc 4320
gatcgccctt cccaacagtt gcgcagcctg aatggcgaat ggcgattccg ttgcaatggc 4380
tggcggtaat attgttctgg atattaccag caaggccgat agtttgagtt cttctactca 4440
ggcaagtgat gttattacta atcaaagaag tattgcgaca acggttaatt tgcgtgatgg 4500
acagactctt ttactcggtg gcctcactga ttataaaaac acttctcagg attctggcgt 4560
accgttcctg tctaaaatcc ctttaatcgg cctcctgttt agctcccgct ctgattctaa 4620
cgaggaaagc acgttatacg tgctcgtcaa agcaaccata gtacgcgccc tgtagcggcg 4680
cattaagcgc ggcgggtgtg gtggttacgc gcagcgtgac cgctacactt gccagcgccc 4740
tagcgcccgc tcctttcgct ttcttccctt cctttctcgc cacgttcgcc ggctttcccc 4800
gtcaagctct aaatcggggg ctccctttag ggttccgatt tagtgcttta cggcacctcg 4860
accccaaaaa acttgattag ggtgatggtt cacgtagtgg gccatcgccc tgatagacgg 4920
tttttcgccc tttgacgttg gagtccacgt tctttaatag tggactcttg ttccaaactg 4980
gaacaacact caaccctatc tcggtctatt cttttgattt ataagggatt ttgccgattt 5040
cggcctattg gttaaaaaat gagctgattt aacaaaaatt taacgcgaat tttaacaaaa 5100
tattaacgtt tacaatttaa atatttgctt atacaatctt cctgtttttg gggcttttct 5160
gattatcaac cggggtacat atgattgaca tgctagtttt acgattaccg ttcatcgatt 5220
ctcttgtttg ctccagactc tcaggcaatg acctgatagc ctttgtagag acctctcaaa 5280
aatagctacc ctctccggca tgaatttatc agctagaacg gttgaatatc atattgatgg 5340
tgatttgact gtctccggcc tttctcaccc gtttgaatct ttacctacac attactcagg 5400
cattgcattt aaaatatatg agggttctaa aaatttttat ccttgcgttg aaataaaggc 5460
ttctcccgca aaagtattac agggtcataa tgtttttggt acaaccgatt tagctttatg 5520
ctctgaggct ttattgctta attttgctaa ttctttgcct tgcctgtatg atttattgga 5580
tgttggaatc gcctgatgcg gtattttctc cttacgcatc tgtgcggtat ttcacaccgc 5640
atatggtgca ctctcagtac aatctgctct gatgccgcat agttaagcca gccccgacac 5700
ccgccaacac ccgctgacgc gccctgacgg gcttgtctgc tcccggcatc cgcttacaga 5760
caagctgtga ccgtctccgg gagctgcatg tgtcagaggt tttcaccgtc atcaccgaaa 5820
cgcgcgagac gaaagggcct cgtgatacgc ctatttttat aggttaatgt catgataata 5880
atggtttctt agacgtcagg tggcactttt cggggaaatg tgcgcggaac ccctatttgt 5940
ttatttttct aaatacattc aaatatgtat ccgctcatga gacaataacc ctgataaatg 6000
cttcaataat attgaaaaag gaagagtatg agtattcaac atttccgtgt cgcccttatt 6060
cccttttttg cggcattttg ccttcctgtt tttgctcacc cagaaacgct ggtgaaagta 6120
aaagatgctg aagatcagtt gggtgcacga gtgggttaca tcgaactgga tctcaacagc 6180
ggtaagatcc ttgagagttt tcgccccgaa gaacgttttc caatgatgag cacttttaaa 6240
gttctgctat gtggcgcggt attatcccgt attgacgccg ggcaagagca actcggtcgc 6300
cgcatacact attctcagaa tgacttggtt gagtactcac cagtcacaga aaagcatctt 6360
acggatggca tgacagtaag agaattatgc agtgctgcca taaccatgag tgataacact 6420
gcggccaact tacttctgac aacgatcgga ggaccgaagg agctaaccgc ttttttgcac 6480
aacatggggg atcatgtaac tcgccttgat cgttgggaac cggagctgaa tgaagccata 6540
ccaaacgacg agcgtgacac cacgatgcct gtagcaatgg caacaacgtt gcgcaaacta 6600
ttaactggcg aactacttac tctagcttcc cggcaacaat taatagactg gatggaggcg 6660
gataaagttg caggaccact tctgcgctcg gcccttccgg ctggctggtt tattgctgat 6720
aaatctggag ccggtgagcg tgggtctcgc ggtatcattg cagcactggg gccagatggt 6780
aagccctccc gtatcgtagt tatctacacg acggggagtc aggcaactat ggatgaacga 6840
aatagacaga tcgctgagat aggtgcctca ctgattaagc attggtaact gtcagaccaa 6900
gtttactcat atatacttta gattgattta aaacttcatt tttaatttaa aaggatctag 6960
gtgaagatcc tttttgataa tctcatgacc aaaatccctt aacgtgagtt ttcgttccac 7020
tgagcgtcag accccgtaga aaagatcaaa ggatcttctt gagatccttt ttttctgcgc 7080
gtaatctgct gcttgcaaac aaaaaaacca ccgctaccag cggtggtttg tttgccggat 7140
caagagctac caactctttt tccgaaggta actggcttca gcagagcgca gataccaaat 7200
actgtccttc tagtgtagcc gtagttaggc caccacttca agaactctgt agcaccgcct 7260
acatacctcg ctctgctaat cctgttacca gtggctgctg ccagtggcga taagtcgtgt 7320
cttaccgggt tggactcaag acgatagtta ccggataagg cgcagcggtc gggctgaacg 7380
gggggttcgt gcacacagcc cagcttggag cgaacgacct acaccgaact gagataccta 7440
cagcgtgagc tatgagaaag cgccacgctt cccgaaggga gaaaggcgga caggtatccg 7500
gtaagcggca gggtcggaac aggagagcgc acgagggagc ttccaggggg aaacgcctgg 7560
tatctttata gtcctgtcgg gtttcgccac ctctgacttg agcgtcgatt tttgtgatgc 7620
tcgtcagggg ggcggagcct atggaaaaac gccagcaacg cggccttttt acggttcctg 7680
gccttttgct ggccttttgc tcacatgttc tttcctgcgt tatcccctga ttctgtggat 7740
aaccgtatta ccgcctttga gtgagctgat accgctcgcc gcagccgaac gaccgagcgc 7800
agcgagtcag tgagcgagga agcggaagag cgcccaatac gcaaaccgcc tctccccgcg 7860
cgttggccga ttcattaatg 7880
<210>52
<211>7593
<212>ДНК
<213>Искусственная последовательность
<220>
<223>Синтезированная конструкция rAAV PD-1 Exon5_MND-BFP_дистальное 5'-
гомологичное плечо
<400>52
cagctgcgcg ctcgctcgct cactgaggcc gcccgggcaa agcccgggcg tcgggcgacc 60
tttggtcgcc cggcctcagt gagcgagcga gcgcgcagag agggagtggc caactccatc 120
actaggggtt ccttgtagtt aatgattaac ccgccatgct acttatctac gtagaattcg 180
gccccactgc ccactgccca gggcagcaat gcccatacca cgtggtccca gctccgagct 240
tgtcctgaaa agggggcaaa gactggaccc tgagcctgcc aaggggccac actcctccca 300
gggctggggt ctccatgggc agccccccac ccacccagac cagttacact cccctgtgcc 360
agagcagtgc agacaggacc aggccaggat gcccaagggt caggggctgg ggatgggtag 420
cccccaaaca gccctttctg ggggactggc ctcaacgggg aagggggtga aggctcttag 480
taggaaatca gggagaccca agtcagagcc aggcgctgtg cagaagctgc agcctcacgt 540
agaaggaaga gcctctgcag tggaggccag tgcccatccc cgggtggcag aggccccagc 600
agagacttct caatgacatt ccagctgggg tggcccttcc agagcccttg ctgcccgagg 660
gatgtgagca ggtggccggg gaggctttgt ggggccaccc agccccttcc tcacctctct 720
ccatctctca gactccccag acaggccctg gaaccccccc accttctccc cagccctgct 780
cgtggtgacc gaaggggaca acgccacctt cacctgcagc ttctccaaca catcggagag 840
cttcgtgcta aactggtacc gcatgagccc cagcaaccag acggacaagc tggccgcctt 900
ccccgaggac cgcagccagc ccggccagga ctgccgcttc cgtgtcacac aactgcccaa 960
cgggcgtgac ttccacatga gcgtggtcag ggcccggcgc aatgacagcg gcacctacct 1020
ctgtggggcc atctccctgg cccccaaggc gcagatcaaa gagagcctgc gggcagagct 1080
cagggtgaca ggtgcggcct cggaggcccc ggggcagggg tgagctgagc cggtcctggg 1140
gtgggtgtcc cctcctgcac aggatcagga gctccagggt cgtagggcag ggacccccca 1200
gctccagtcc agggctctgt cctgcacctg gggaatggtg accggcatct ctgtcctcta 1260
gctctggaag caccccagcc cctctagtct gccctcaccc ctgaccctga ccctccaccc 1320
tgaccccgtc ctaacccctg acctttgtgc ccttccagag agaagggcag aagtgcccac 1380
agcccacccc agcccctcac ccaggccagc cggccagttc caaaccctgg tggttggtgt 1440
cgtgggcggc ctgctgggca gcctggtgct gctagtctgg gtcctggccg tcatctatgc 1500
atgaacagag aaacaggaga atatgggcca aacaggatat ctgtggtaag cagttcctgc 1560
cccggctcag ggccaagaac agttggaaca gcagaatatg ggccaaacag gatatctgtg 1620
gtaagcagtt cctgccccgg ctcagggcca agaacagatg gtccccagat gcggtcccgc 1680
cctcagcagt ttctagagaa ccatcagatg tttccagggt gccccaagga cctgaaatga 1740
ccctgtgcct tatttgaact aaccaatcag ttcgcttctc gcttctgttc gcgcgcttct 1800
gctccccgag ctctatataa gcagagctcg tttagtgaac cgtcagatcg cctggagacg 1860
ccatccacgc tgttttgact tccatagaag gatctcgagg ccaccatggc tagcgagctg 1920
attaaggaga acatgcacat gaagctgtac atggagggca ccgtggacaa ccatcacttc 1980
aagtgcacat ccgagggcga aggcaagccc tacgagggca cccagaccat gagaatcaag 2040
gtggtcgagg gcggccctct ccccttcgcc ttcgacatcc tggctactag cttcctctac 2100
ggcagcaaga ccttcatcaa ccacacccag ggcatccccg acttcttcaa gcagtccttc 2160
cctgagggct tcacatggga gagagtcacc acatacgagg acgggggcgt gctgaccgct 2220
acccaggaca ccagcctcca ggacggctgc ctcatctaca acgtcaagat cagaggggtg 2280
aacttcacat ccaacggccc tgtgatgcag aagaaaacac tcggctggga ggccttcacc 2340
gagacgctgt accccgctga cggcggcctg gaaggcagaa acgacatggc cctgaagctc 2400
gtgggcggga gccatctgat cgcaaacatc aagaccacat atagatccaa gaaacccgct 2460
aagaacctca agatgcctgg cgtctactat gtggactaca gactggaaag aatcaaggag 2520
gccaacaacg agacctacgt cgagcagcac gaggtggcag tggccagata ctgcgacctc 2580
cctagcaaac tggggcacaa gctaaattga aagctttgct ttatttgtga aatttgtgat 2640
gctattgctt tatttgtaac cattataagc tgcaataaac aagtttaaca acaacaattg 2700
cattcatttt atgtttcagg ttcaggggga ggtgtgggag gttttttaaa gtcgacccac 2760
cattgtcttt cctagcggaa tgggcacctc atcccccgcc cgcaggggct cagctgacgg 2820
ccctcggagt gcccagccac tgaggcctga ggatggacac tgctcttggc ccctctgacc 2880
ggcttccttg gccaccagtg ttctgcagac cctccaccat gagcccgggt cagcgcattt 2940
cctcaggaga agcaggcagg gtgcaggcca ttgcaggccg tccaggggct gagctgcctg 3000
ggggcgaccg gggctccagc ctgcacctgc accaggcaca gccccaccac aggactcatg 3060
tctcaatgcc cacagtgagc ccaggcagca ggtgtcaccg tcccctacag ggagggccag 3120
atgcagtcac tgcttcaggt cctgccagca cagagctgcc tgcgtccagc tccctgaatc 3180
tctgctgctg ctgctgctgc tgctgctgct gcctgcggcc cggggctgaa ggcgccgtgg 3240
ccctgcctga cgccccggag cctcctgcct gaacttgggg gctggttgga gatggccttg 3300
gagcagccaa ggtgcccctg gcagtggcat cccgaaacgc cctggacgca gggcccaaga 3360
ctgggcacag gagtgggagg tacatggggc tggggactcc ccaggagtta tctgctccct 3420
gcaggcctag agaagtttca gggaaggtca gaagagctcc tggctgtggt gggcagggca 3480
ggaaacccct ccacctttac acatgcccag gcagcacctc aggccctttg tggggcaggg 3540
aagctgaggc agtaagcggg caggcagagc tggaggcctt tcaggcccag ccagcactct 3600
ggcctcctgc cgccgcattc caccccagcc cctcacacca ctcgggagag ggacatccta 3660
cggtcccaag gtcaggaggg cagggctggg gttgactcag gcccctccca gctgtggcca 3720
cctgggtgtt gggagggcag aagtgcaggc acctagggcc ccccatgtgc ccaccctggg 3780
agctctcctt ggaacccatt cctgaaatta tttaaagggg ttggccggac tagttacgta 3840
gataagtagc atggcgggtt aatcattaac tacaaggaac ccctagtgat ggagttggcc 3900
actccctctc tgcgcgctcg ctcgctcact gaggccgggc gaccaaaggt cgcccgacgc 3960
ccgggctttg cccgggcggc ctcagtgagc gagcgagcgc gccagctggc gtaatagcga 4020
agaggcccgc accgatcgcc cttcccaaca gttgcgcagc ctgaatggcg aatggcgatt 4080
ccgttgcaat ggctggcggt aatattgttc tggatattac cagcaaggcc gatagtttga 4140
gttcttctac tcaggcaagt gatgttatta ctaatcaaag aagtattgcg acaacggtta 4200
atttgcgtga tggacagact cttttactcg gtggcctcac tgattataaa aacacttctc 4260
aggattctgg cgtaccgttc ctgtctaaaa tccctttaat cggcctcctg tttagctccc 4320
gctctgattc taacgaggaa agcacgttat acgtgctcgt caaagcaacc atagtacgcg 4380
ccctgtagcg gcgcattaag cgcggcgggt gtggtggtta cgcgcagcgt gaccgctaca 4440
cttgccagcg ccctagcgcc cgctcctttc gctttcttcc cttcctttct cgccacgttc 4500
gccggctttc cccgtcaagc tctaaatcgg gggctccctt tagggttccg atttagtgct 4560
ttacggcacc tcgaccccaa aaaacttgat tagggtgatg gttcacgtag tgggccatcg 4620
ccctgataga cggtttttcg ccctttgacg ttggagtcca cgttctttaa tagtggactc 4680
ttgttccaaa ctggaacaac actcaaccct atctcggtct attcttttga tttataaggg 4740
attttgccga tttcggccta ttggttaaaa aatgagctga tttaacaaaa atttaacgcg 4800
aattttaaca aaatattaac gtttacaatt taaatatttg cttatacaat cttcctgttt 4860
ttggggcttt tctgattatc aaccggggta catatgattg acatgctagt tttacgatta 4920
ccgttcatcg attctcttgt ttgctccaga ctctcaggca atgacctgat agcctttgta 4980
gagacctctc aaaaatagct accctctccg gcatgaattt atcagctaga acggttgaat 5040
atcatattga tggtgatttg actgtctccg gcctttctca cccgtttgaa tctttaccta 5100
cacattactc aggcattgca tttaaaatat atgagggttc taaaaatttt tatccttgcg 5160
ttgaaataaa ggcttctccc gcaaaagtat tacagggtca taatgttttt ggtacaaccg 5220
atttagcttt atgctctgag gctttattgc ttaattttgc taattctttg ccttgcctgt 5280
atgatttatt ggatgttgga atcgcctgat gcggtatttt ctccttacgc atctgtgcgg 5340
tatttcacac cgcatatggt gcactctcag tacaatctgc tctgatgccg catagttaag 5400
ccagccccga cacccgccaa cacccgctga cgcgccctga cgggcttgtc tgctcccggc 5460
atccgcttac agacaagctg tgaccgtctc cgggagctgc atgtgtcaga ggttttcacc 5520
gtcatcaccg aaacgcgcga gacgaaaggg cctcgtgata cgcctatttt tataggttaa 5580
tgtcatgata ataatggttt cttagacgtc aggtggcact tttcggggaa atgtgcgcgg 5640
aacccctatt tgtttatttt tctaaataca ttcaaatatg tatccgctca tgagacaata 5700
accctgataa atgcttcaat aatattgaaa aaggaagagt atgagtattc aacatttccg 5760
tgtcgccctt attccctttt ttgcggcatt ttgccttcct gtttttgctc acccagaaac 5820
gctggtgaaa gtaaaagatg ctgaagatca gttgggtgca cgagtgggtt acatcgaact 5880
ggatctcaac agcggtaaga tccttgagag ttttcgcccc gaagaacgtt ttccaatgat 5940
gagcactttt aaagttctgc tatgtggcgc ggtattatcc cgtattgacg ccgggcaaga 6000
gcaactcggt cgccgcatac actattctca gaatgacttg gttgagtact caccagtcac 6060
agaaaagcat cttacggatg gcatgacagt aagagaatta tgcagtgctg ccataaccat 6120
gagtgataac actgcggcca acttacttct gacaacgatc ggaggaccga aggagctaac 6180
cgcttttttg cacaacatgg gggatcatgt aactcgcctt gatcgttggg aaccggagct 6240
gaatgaagcc ataccaaacg acgagcgtga caccacgatg cctgtagcaa tggcaacaac 6300
gttgcgcaaa ctattaactg gcgaactact tactctagct tcccggcaac aattaataga 6360
ctggatggag gcggataaag ttgcaggacc acttctgcgc tcggcccttc cggctggctg 6420
gtttattgct gataaatctg gagccggtga gcgtgggtct cgcggtatca ttgcagcact 6480
ggggccagat ggtaagccct cccgtatcgt agttatctac acgacgggga gtcaggcaac 6540
tatggatgaa cgaaatagac agatcgctga gataggtgcc tcactgatta agcattggta 6600
actgtcagac caagtttact catatatact ttagattgat ttaaaacttc atttttaatt 6660
taaaaggatc taggtgaaga tcctttttga taatctcatg accaaaatcc cttaacgtga 6720
gttttcgttc cactgagcgt cagaccccgt agaaaagatc aaaggatctt cttgagatcc 6780
tttttttctg cgcgtaatct gctgcttgca aacaaaaaaa ccaccgctac cagcggtggt 6840
ttgtttgccg gatcaagagc taccaactct ttttccgaag gtaactggct tcagcagagc 6900
gcagatacca aatactgtcc ttctagtgta gccgtagtta ggccaccact tcaagaactc 6960
tgtagcaccg cctacatacc tcgctctgct aatcctgtta ccagtggctg ctgccagtgg 7020
cgataagtcg tgtcttaccg ggttggactc aagacgatag ttaccggata aggcgcagcg 7080
gtcgggctga acggggggtt cgtgcacaca gcccagcttg gagcgaacga cctacaccga 7140
actgagatac ctacagcgtg agctatgaga aagcgccacg cttcccgaag ggagaaaggc 7200
ggacaggtat ccggtaagcg gcagggtcgg aacaggagag cgcacgaggg agcttccagg 7260
gggaaacgcc tggtatcttt atagtcctgt cgggtttcgc cacctctgac ttgagcgtcg 7320
atttttgtga tgctcgtcag gggggcggag cctatggaaa aacgccagca acgcggcctt 7380
tttacggttc ctggcctttt gctggccttt tgctcacatg ttctttcctg cgttatcccc 7440
tgattctgtg gataaccgta ttaccgcctt tgagtgagct gataccgctc gccgcagccg 7500
aacgaccgag cgcagcgagt cagtgagcga ggaagcggaa gagcgcccaa tacgcaaacc 7560
gcctctcccc gcgcgttggc cgattcatta atg 7593
<210>53
<211>6718
<212>ДНК
<213>Искусственная последовательность
<220>
<223>Синтезированная конструкция rAAV PD-1 Exon1_MND-GFP
<400>53
cagctgcgcg ctcgctcgct cactgaggcc gcccgggcaa agcccgggcg tcgggcgacc 60
tttggtcgcc cggcctcagt gagcgagcga gcgcgcagag agggagtggc caactccatc 120
actaggggtt ccttgtagtt aatgattaac ccgccatgct acttatctac gtaaggctgt 180
tgcaggcatc acacggtgga aagatctgga actgtggcca tggtgtgagg ccatccacaa 240
ggtggaagct ttgaggggga gccgattagc catggacagt tgtcattcag tagggtcacc 300
tgtgccccag cgaaggggga tgggccggga aggcagaggc caggcacctg cccccagcag 360
gggcagaggc tgtgggcagc cgggaggctc ccagaggctc cgacagaatg ggagtggggt 420
tgagcccacc cctcactgca gcccaggaac ctgagcccag agggggccac ccaccttccc 480
caggcaggga ggcccggccc ccagggagat gggggggatg ggggaggaga agggcctgcc 540
cccacccggc agcctcagga ggggcagctc gggcgggata tggaaagagg ccacagcagt 600
gagcagagac acagaggagg aaggggccct gagctgggga gacccccacg gggtagggcg 660
tgggggccac gggcccacct cctccccatc tcctctgtct ccctgtctct gtctctctct 720
ccctccccca ccctctcccc agtcctaccc cctcctcacc cctcctcccc cagcactgcc 780
tctgtcactc tcgcccacgt ggatgtggag gaagaggggg cgggagcaag gggcgggcac 840
cctcccttca acctgacctg ggacagtttc ccttccgctc acctccgcct gagcagtgga 900
gaaggcggca ctctggtggg gctgctccaa cgcgtgaaca gagaaacagg agaatatggg 960
ccaaacagga tatctgtggt aagcagttcc tgccccggct cagggccaag aacagttgga 1020
acagcagaat atgggccaaa caggatatct gtggtaagca gttcctgccc cggctcaggg 1080
ccaagaacag atggtcccca gatgcggtcc cgccctcagc agtttctaga gaaccatcag 1140
atgtttccag ggtgccccaa ggacctgaaa tgaccctgtg ccttatttga actaaccaat 1200
cagttcgctt ctcgcttctg ttcgcgcgct tctgctcccc gagctctata taagcagagc 1260
tcgtttagtg aaccgtcaga tcgcctggag acgccatcca cgctgttttg acttccatag 1320
aaggatctcg aggccaccat ggtgagcaag ggcgaggagc tgttcaccgg ggtggtgccc 1380
atcctggtcg agctggacgg cgacgtaaac ggccacaagt tcagcgtgtc cggcgagggc 1440
gagggcgatg ccacctacgg caagctgacc ctgaagttca tctgcaccac cggcaagctg 1500
cccgtgccct ggcccaccct cgtgaccacc ctgacctacg gcgtgcagtg cttcagccgc 1560
taccccgacc acatgaagca gcacgacttc ttcaagtccg ccatgcccga aggctacgtc 1620
caggagcgca ccatcttctt caaggacgac ggcaactaca agacccgcgc cgaggtgaag 1680
ttcgagggcg acaccctggt gaaccgcatc gagctgaagg gcatcgactt caaggaggac 1740
ggcaacatcc tggggcacaa gctggagtac aactacaaca gccacaacgt ctatatcatg 1800
gccgacaagc agaagaacgg catcaaggtg aacttcaaga tccgccacaa catcgaggac 1860
ggcagcgtgc agctcgccga ccactaccag cagaacaccc ccatcggcga cggccccgtg 1920
ctgctgcccg acaaccacta cctgagcacc cagtccgccc tgagcaaaga ccccaacgag 1980
aagcgcgatc acatggtcct gctggagttc gtgaccgccg ccgggatcac tctcggcatg 2040
gacgagctgt acaagtaagc ggccgcgctt tatttgtgaa atttgtgatg ctattgcttt 2100
atttgtaacc attataagct gcaataaaca agttaacaac aacaattgca ttcattttat 2160
gtttcaggtt cagggggaga tgtgggaggt tttttaaagc ctcaccggtt ctgggcggtg 2220
ctacaactgg gctggcggcc aggatggttc ttaggtaggt ggggtcggcg gtcaggtgtc 2280
ccagagccag gggtctggag ggaccttcca ccctcagtcc ctggcaggtc ggggggtgct 2340
gaggcgggcc tggccctggc agcccagggg tcccggagcg aggggtctgg agggaccttt 2400
cactctcagt ccctggcagg tcggggggtg ctgtggcagg cccagccttg gcccccagct 2460
ctgcccctta ccctgagctg tgtggctttg ggcagctcga actcctgggt tcctctctgg 2520
gccccaactc ctcccctggc ccaagtcccc tctttgctcc tgggcaggca ggacctctgt 2580
cccctctcag ccggtccttg gggctgcgtg tttctgtaga atgacgggtc aggctggcca 2640
gaaccccaaa ccttggccgt ggggagtctg cgtggcggct ctgccttgcc caggcatcct 2700
tggtcctcac tcgagttttc ctaaggatgg gatgagcccc atgtgggact aaccttggct 2760
ttacgacgtc aaagtttaga tgagctggtg atatttttct cattatatcc aaagtgtacc 2820
tgttcgagtg aggacagttc ttctgtctcc aggatccctc ctgggtgggg attgtgcccg 2880
cctgggtctc tgcccagatt ccagggctct ccccgagccc tgttcagacc atccgtgggg 2940
gaggccttgg cctcactctt acgtagataa gtagcatggc gggttaatca ttaactacaa 3000
ggaaccccta gtgatggagt tggccactcc ctctctgcgc gctcgctcgc tcactgaggc 3060
cgggcgacca aaggtcgccc gacgcccggg ctttgcccgg gcggcctcag tgagcgagcg 3120
agcgcgccag ctggcgtaat agcgaagagg cccgcaccga tcgcccttcc caacagttgc 3180
gcagcctgaa tggcgaatgg cgattccgtt gcaatggctg gcggtaatat tgttctggat 3240
attaccagca aggccgatag tttgagttct tctactcagg caagtgatgt tattactaat 3300
caaagaagta ttgcgacaac ggttaatttg cgtgatggac agactctttt actcggtggc 3360
ctcactgatt ataaaaacac ttctcaggat tctggcgtac cgttcctgtc taaaatccct 3420
ttaatcggcc tcctgtttag ctcccgctct gattctaacg aggaaagcac gttatacgtg 3480
ctcgtcaaag caaccatagt acgcgccctg tagcggcgca ttaagcgcgg cgggtgtggt 3540
ggttacgcgc agcgtgaccg ctacacttgc cagcgcccta gcgcccgctc ctttcgcttt 3600
cttcccttcc tttctcgcca cgttcgccgg ctttccccgt caagctctaa atcgggggct 3660
ccctttaggg ttccgattta gtgctttacg gcacctcgac cccaaaaaac ttgattaggg 3720
tgatggttca cgtagtgggc catcgccctg atagacggtt tttcgccctt tgacgttgga 3780
gtccacgttc tttaatagtg gactcttgtt ccaaactgga acaacactca accctatctc 3840
ggtctattct tttgatttat aagggatttt gccgatttcg gcctattggt taaaaaatga 3900
gctgatttaa caaaaattta acgcgaattt taacaaaata ttaacgttta caatttaaat 3960
atttgcttat acaatcttcc tgtttttggg gcttttctga ttatcaaccg gggtacatat 4020
gattgacatg ctagttttac gattaccgtt catcgattct cttgtttgct ccagactctc 4080
aggcaatgac ctgatagcct ttgtagagac ctctcaaaaa tagctaccct ctccggcatg 4140
aatttatcag ctagaacggt tgaatatcat attgatggtg atttgactgt ctccggcctt 4200
tctcacccgt ttgaatcttt acctacacat tactcaggca ttgcatttaa aatatatgag 4260
ggttctaaaa atttttatcc ttgcgttgaa ataaaggctt ctcccgcaaa agtattacag 4320
ggtcataatg tttttggtac aaccgattta gctttatgct ctgaggcttt attgcttaat 4380
tttgctaatt ctttgccttg cctgtatgat ttattggatg ttggaatcgc ctgatgcggt 4440
attttctcct tacgcatctg tgcggtattt cacaccgcat atggtgcact ctcagtacaa 4500
tctgctctga tgccgcatag ttaagccagc cccgacaccc gccaacaccc gctgacgcgc 4560
cctgacgggc ttgtctgctc ccggcatccg cttacagaca agctgtgacc gtctccggga 4620
gctgcatgtg tcagaggttt tcaccgtcat caccgaaacg cgcgagacga aagggcctcg 4680
tgatacgcct atttttatag gttaatgtca tgataataat ggtttcttag acgtcaggtg 4740
gcacttttcg gggaaatgtg cgcggaaccc ctatttgttt atttttctaa atacattcaa 4800
atatgtatcc gctcatgaga caataaccct gataaatgct tcaataatat tgaaaaagga 4860
agagtatgag tattcaacat ttccgtgtcg cccttattcc cttttttgcg gcattttgcc 4920
ttcctgtttt tgctcaccca gaaacgctgg tgaaagtaaa agatgctgaa gatcagttgg 4980
gtgcacgagt gggttacatc gaactggatc tcaacagcgg taagatcctt gagagttttc 5040
gccccgaaga acgttttcca atgatgagca cttttaaagt tctgctatgt ggcgcggtat 5100
tatcccgtat tgacgccggg caagagcaac tcggtcgccg catacactat tctcagaatg 5160
acttggttga gtactcacca gtcacagaaa agcatcttac ggatggcatg acagtaagag 5220
aattatgcag tgctgccata accatgagtg ataacactgc ggccaactta cttctgacaa 5280
cgatcggagg accgaaggag ctaaccgctt ttttgcacaa catgggggat catgtaactc 5340
gccttgatcg ttgggaaccg gagctgaatg aagccatacc aaacgacgag cgtgacacca 5400
cgatgcctgt agcaatggca acaacgttgc gcaaactatt aactggcgaa ctacttactc 5460
tagcttcccg gcaacaatta atagactgga tggaggcgga taaagttgca ggaccacttc 5520
tgcgctcggc ccttccggct ggctggttta ttgctgataa atctggagcc ggtgagcgtg 5580
ggtctcgcgg tatcattgca gcactggggc cagatggtaa gccctcccgt atcgtagtta 5640
tctacacgac ggggagtcag gcaactatgg atgaacgaaa tagacagatc gctgagatag 5700
gtgcctcact gattaagcat tggtaactgt cagaccaagt ttactcatat atactttaga 5760
ttgatttaaa acttcatttt taatttaaaa ggatctaggt gaagatcctt tttgataatc 5820
tcatgaccaa aatcccttaa cgtgagtttt cgttccactg agcgtcagac cccgtagaaa 5880
agatcaaagg atcttcttga gatccttttt ttctgcgcgt aatctgctgc ttgcaaacaa 5940
aaaaaccacc gctaccagcg gtggtttgtt tgccggatca agagctacca actctttttc 6000
cgaaggtaac tggcttcagc agagcgcaga taccaaatac tgtccttcta gtgtagccgt 6060
agttaggcca ccacttcaag aactctgtag caccgcctac atacctcgct ctgctaatcc 6120
tgttaccagt ggctgctgcc agtggcgata agtcgtgtct taccgggttg gactcaagac 6180
gatagttacc ggataaggcg cagcggtcgg gctgaacggg gggttcgtgc acacagccca 6240
gcttggagcg aacgacctac accgaactga gatacctaca gcgtgagcta tgagaaagcg 6300
ccacgcttcc cgaagggaga aaggcggaca ggtatccggt aagcggcagg gtcggaacag 6360
gagagcgcac gagggagctt ccagggggaa acgcctggta tctttatagt cctgtcgggt 6420
ttcgccacct ctgacttgag cgtcgatttt tgtgatgctc gtcagggggg cggagcctat 6480
ggaaaaacgc cagcaacgcg gcctttttac ggttcctggc cttttgctgg ccttttgctc 6540
acatgttctt tcctgcgtta tcccctgatt ctgtggataa ccgtattacc gcctttgagt 6600
gagctgatac cgctcgccgc agccgaacga ccgagcgcag cgagtcagtg agcgaggaag 6660
cggaagagcg cccaatacgc aaaccgcctc tccccgcgcg ttggccgatt cattaatg 6718
<210>54
<211>756
<212>ДНК
<213>Homo sapiens
<400>54
aggctgttgc aggcatcaca cggtggaaag atctggaact gtggccatgg tgtgaggcca 60
tccacaaggt ggaagctttg agggggagcc gattagccat ggacagttgt cattcagtag 120
ggtcacctgt gccccagcga agggggatgg gccgggaagg cagaggccag gcacctgccc 180
ccagcagggg cagaggctgt gggcagccgg gaggctccca gaggctccga cagaatggga 240
gtggggttga gcccacccct cactgcagcc caggaacctg agcccagagg gggccaccca 300
ccttccccag gcagggaggc ccggccccca gggagatggg ggggatgggg gaggagaagg 360
gcctgccccc acccggcagc ctcaggaggg gcagctcggg cgggatatgg aaagaggcca 420
cagcagtgag cagagacaca gaggaggaag gggccctgag ctggggagac ccccacgggg 480
tagggcgtgg gggccacggg cccacctcct ccccatctcc tctgtctccc tgtctctgtc 540
tctctctccc tcccccaccc tctccccagt cctaccccct cctcacccct cctcccccag 600
cactgcctct gtcactctcg cccacgtgga tgtggaggaa gagggggcgg gagcaagggg 660
cgggcaccct cccttcaacc tgacctggga cagtttccct tccgctcacc tccgcctgag 720
cagtggagaa ggcggcactc tggtggggct gctcca 756
<210>55
<211>750
<212>ДНК
<213>Homo sapiens
<400>55
tctgggcggt gctacaactg ggctggcggc caggatggtt cttaggtagg tggggtcggc 60
ggtcaggtgt cccagagcca ggggtctgga gggaccttcc accctcagtc cctggcaggt 120
cggggggtgc tgaggcgggc ctggccctgg cagcccaggg gtcccggagc gaggggtctg 180
gagggacctt tcactctcag tccctggcag gtcggggggt gctgtggcag gcccagcctt 240
ggcccccagc tctgcccctt accctgagct gtgtggcttt gggcagctcg aactcctggg 300
ttcctctctg ggccccaact cctcccctgg cccaagtccc ctctttgctc ctgggcaggc 360
aggacctctg tcccctctca gccggtcctt ggggctgcgt gtttctgtag aatgacgggt 420
caggctggcc agaaccccaa accttggccg tggggagtct gcgtggcggc tctgccttgc 480
ccaggcatcc ttggtcctca ctcgagtttt cctaaggatg ggatgagccc catgtgggac 540
taaccttggc tttacgacgt caaagtttag atgagctggt gatatttttc tcattatatc 600
caaagtgtac ctgttcgagt gaggacagtt cttctgtctc caggatccct cctgggtggg 660
gattgtgccc gcctgggtct ctgcccagat tccagggctc tccccgagcc ctgttcagac 720
catccgtggg ggaggccttg gcctcactct 750
<210>56
<211>7609
<212>ДНК
<213>Искусственная последовательность
<220>
<223>Синтезированная конструкция rAAV PD-1 Exon1_MND-CD19CAR
<400>56
cagctgcgcg ctcgctcgct cactgaggcc gcccgggcaa agcccgggcg tcgggcgacc 60
tttggtcgcc cggcctcagt gagcgagcga gcgcgcagag agggagtggc caactccatc 120
actaggggtt ccttgtagtt aatgattaac ccgccatgct acttatctac gtaaggctgt 180
tgcaggcatc acacggtgga aagatctgga actgtggcca tggtgtgagg ccatccacaa 240
ggtggaagct ttgaggggga gccgattagc catggacagt tgtcattcag tagggtcacc 300
tgtgccccag cgaaggggga tgggccggga aggcagaggc caggcacctg cccccagcag 360
gggcagaggc tgtgggcagc cgggaggctc ccagaggctc cgacagaatg ggagtggggt 420
tgagcccacc cctcactgca gcccaggaac ctgagcccag agggggccac ccaccttccc 480
caggcaggga ggcccggccc ccagggagat gggggggatg ggggaggaga agggcctgcc 540
cccacccggc agcctcagga ggggcagctc gggcgggata tggaaagagg ccacagcagt 600
gagcagagac acagaggagg aaggggccct gagctgggga gacccccacg gggtagggcg 660
tgggggccac gggcccacct cctccccatc tcctctgtct ccctgtctct gtctctctct 720
ccctccccca ccctctcccc agtcctaccc cctcctcacc cctcctcccc cagcactgcc 780
tctgtcactc tcgcccacgt ggatgtggag gaagaggggg cgggagcaag gggcgggcac 840
cctcccttca acctgacctg ggacagtttc ccttccgctc acctccgcct gagcagtgga 900
gaaggcggca ctctggtggg gctgctccaa cgcgtgatcc atcgattagt ccaatttgtt 960
aaagacagga tatcagtggt ccaggctcta gttttgactc aacaatatca ccagctgaag 1020
cctatagagt acgagccata gatagaataa aagattttat ttagtctcca gaaaaagggg 1080
ggaatgaaag accccacctg taggtttggc aagctaggat caaggttagg aacagagaga 1140
cagcagaata tgggccaaac aggatatctg tggtaagcag ttcctgcccc ggctcagggc 1200
caagaacagt tggaacagca gaatatgggc caaacaggat atctgtggta agcagttcct 1260
gccccggctc agggccaaga acagatggtc cccagatgcg gtcccgccct cagcagtttc 1320
tagagaacca tcagatgttt ccagggtgcc ccaaggacct gaaatgaccc tgtgccttat 1380
ttgaactaac caatcagttc gcttctcgct tctgttcgcg cgcttctgct ccccgagctc 1440
aataaaagag cccacaaccc ctcactcggc gcgacgcgtc atagccacca tggccttacc 1500
agtgaccgcc ttgctcctgc cgctggcctt gctgctccac gccgccaggc cggacatcca 1560
gatgacacag actacatcct ccctgtctgc ctctctggga gacagagtca ccatcagttg 1620
cagggcaagt caggacatta gtaaatattt aaattggtat cagcagaaac cagatggaac 1680
tgttaaactc ctgatctacc atacatcaag attacactca ggagtcccat caaggttcag 1740
tggcagtggg tctggaacag attattctct caccattagc aacctggagc aagaagatat 1800
tgccacttac ttttgccaac agggtaatac gcttccgtac acgttcggag gggggaccaa 1860
gctggagatc acaggtggcg gtggctccgg cggtggtggg tctggtggcg gcggaagcga 1920
ggtgaaactg caggagtcag gacctggcct ggtggcgccc tcacagagcc tgtccgtcac 1980
atgcactgtc tcaggggtct cattacccga ctatggtgta agctggattc gccagcctcc 2040
acgaaagggt ctggagtggc tgggagtaat atggggtagt gaaaccacat actataattc 2100
agctctcaaa tccagactga ccatcatcaa ggacaactcc aagagccaag ttttcttaaa 2160
aatgaacagt ctgcaaactg atgacacagc catttactac tgtgccaaac attattacta 2220
cggtggtagc tatgctatgg actactgggg tcaaggaacc tcggtcaccg tctcctcaac 2280
cacgacgcca gcgccgcgac caccaacacc ggcgcccacc atcgcgtcgc agcccctgtc 2340
cctgcgccca gaggcgtgcc ggccagcggc ggggggcgca gtgcacacga gggggctgga 2400
cttcgcctgt gatatctaca tctgggcgcc cttggccggg acttgtgggg tccttctcct 2460
gtcactggtg atcacccttt actgcaaacg gggcagaaag aaactcctgt atatattcaa 2520
acaaccattt atgagaccag tacaaactac tcaagaggaa gatggctgta gctgccgatt 2580
tccagaagaa gaagaaggag gatgtgaact gagagtgaag ttcagcagga gcgcagacgc 2640
ccccgcgtac cagcagggcc agaaccagct ctataacgag ctcaatctag gacgaagaga 2700
ggagtacgat gttttggaca agagacgtgg ccgggaccct gagatggggg gaaagccgag 2760
aaggaagaac cctcaggaag gcctgtacaa tgaactgcag aaagataaga tggcggaggc 2820
ctacagtgag attgggatga aaggcgagcg ccggaggggc aaggggcacg atggccttta 2880
ccagggtctc agtacagcca ccaaggacac ctacgacgcc cttcacatgc aggccctgcc 2940
ccctcgctaa gcggccgcgc tttatttgtg aaatttgtga tgctattgct ttatttgtaa 3000
ccattataag ctgcaataaa caagttaaca acaacaattg cattcatttt atgtttcagg 3060
ttcaggggga gatgtgggag gttttttaaa gctcaccggt tctgggcggt gctacaactg 3120
ggctggcggc caggatggtt cttaggtagg tggggtcggc ggtcaggtgt cccagagcca 3180
ggggtctgga gggaccttcc accctcagtc cctggcaggt cggggggtgc tgaggcgggc 3240
ctggccctgg cagcccaggg gtcccggagc gaggggtctg gagggacctt tcactctcag 3300
tccctggcag gtcggggggt gctgtggcag gcccagcctt ggcccccagc tctgcccctt 3360
accctgagct gtgtggcttt gggcagctcg aactcctggg ttcctctctg ggccccaact 3420
cctcccctgg cccaagtccc ctctttgctc ctgggcaggc aggacctctg tcccctctca 3480
gccggtcctt ggggctgcgt gtttctgtag aatgacgggt caggctggcc agaaccccaa 3540
accttggccg tggggagtct gcgtggcggc tctgccttgc ccaggcatcc ttggtcctca 3600
ctcgagtttt cctaaggatg ggatgagccc catgtgggac taaccttggc tttacgacgt 3660
caaagtttag atgagctggt gatatttttc tcattatatc caaagtgtac ctgttcgagt 3720
gaggacagtt cttctgtctc caggatccct cctgggtggg gattgtgccc gcctgggtct 3780
ctgcccagat tccagggctc tccccgagcc ctgttcagac catccgtggg ggaggccttg 3840
gcctcactct tacgtagata agtagcatgg cgggttaatc attaactaca aggaacccct 3900
agtgatggag ttggccactc cctctctgcg cgctcgctcg ctcactgagg ccgggcgacc 3960
aaaggtcgcc cgacgcccgg gctttgcccg ggcggcctca gtgagcgagc gagcgcgcca 4020
gctggcgtaa tagcgaagag gcccgcaccg atcgcccttc ccaacagttg cgcagcctga 4080
atggcgaatg gcgattccgt tgcaatggct ggcggtaata ttgttctgga tattaccagc 4140
aaggccgata gtttgagttc ttctactcag gcaagtgatg ttattactaa tcaaagaagt 4200
attgcgacaa cggttaattt gcgtgatgga cagactcttt tactcggtgg cctcactgat 4260
tataaaaaca cttctcagga ttctggcgta ccgttcctgt ctaaaatccc tttaatcggc 4320
ctcctgttta gctcccgctc tgattctaac gaggaaagca cgttatacgt gctcgtcaaa 4380
gcaaccatag tacgcgccct gtagcggcgc attaagcgcg gcgggtgtgg tggttacgcg 4440
cagcgtgacc gctacacttg ccagcgccct agcgcccgct cctttcgctt tcttcccttc 4500
ctttctcgcc acgttcgccg gctttccccg tcaagctcta aatcgggggc tccctttagg 4560
gttccgattt agtgctttac ggcacctcga ccccaaaaaa cttgattagg gtgatggttc 4620
acgtagtggg ccatcgccct gatagacggt ttttcgccct ttgacgttgg agtccacgtt 4680
ctttaatagt ggactcttgt tccaaactgg aacaacactc aaccctatct cggtctattc 4740
ttttgattta taagggattt tgccgatttc ggcctattgg ttaaaaaatg agctgattta 4800
acaaaaattt aacgcgaatt ttaacaaaat attaacgttt acaatttaaa tatttgctta 4860
tacaatcttc ctgtttttgg ggcttttctg attatcaacc ggggtacata tgattgacat 4920
gctagtttta cgattaccgt tcatcgattc tcttgtttgc tccagactct caggcaatga 4980
cctgatagcc tttgtagaga cctctcaaaa atagctaccc tctccggcat gaatttatca 5040
gctagaacgg ttgaatatca tattgatggt gatttgactg tctccggcct ttctcacccg 5100
tttgaatctt tacctacaca ttactcaggc attgcattta aaatatatga gggttctaaa 5160
aatttttatc cttgcgttga aataaaggct tctcccgcaa aagtattaca gggtcataat 5220
gtttttggta caaccgattt agctttatgc tctgaggctt tattgcttaa ttttgctaat 5280
tctttgcctt gcctgtatga tttattggat gttggaatcg cctgatgcgg tattttctcc 5340
ttacgcatct gtgcggtatt tcacaccgca tatggtgcac tctcagtaca atctgctctg 5400
atgccgcata gttaagccag ccccgacacc cgccaacacc cgctgacgcg ccctgacggg 5460
cttgtctgct cccggcatcc gcttacagac aagctgtgac cgtctccggg agctgcatgt 5520
gtcagaggtt ttcaccgtca tcaccgaaac gcgcgagacg aaagggcctc gtgatacgcc 5580
tatttttata ggttaatgtc atgataataa tggtttctta gacgtcaggt ggcacttttc 5640
ggggaaatgt gcgcggaacc cctatttgtt tatttttcta aatacattca aatatgtatc 5700
cgctcatgag acaataaccc tgataaatgc ttcaataata ttgaaaaagg aagagtatga 5760
gtattcaaca tttccgtgtc gcccttattc ccttttttgc ggcattttgc cttcctgttt 5820
ttgctcaccc agaaacgctg gtgaaagtaa aagatgctga agatcagttg ggtgcacgag 5880
tgggttacat cgaactggat ctcaacagcg gtaagatcct tgagagtttt cgccccgaag 5940
aacgttttcc aatgatgagc acttttaaag ttctgctatg tggcgcggta ttatcccgta 6000
ttgacgccgg gcaagagcaa ctcggtcgcc gcatacacta ttctcagaat gacttggttg 6060
agtactcacc agtcacagaa aagcatctta cggatggcat gacagtaaga gaattatgca 6120
gtgctgccat aaccatgagt gataacactg cggccaactt acttctgaca acgatcggag 6180
gaccgaagga gctaaccgct tttttgcaca acatggggga tcatgtaact cgccttgatc 6240
gttgggaacc ggagctgaat gaagccatac caaacgacga gcgtgacacc acgatgcctg 6300
tagcaatggc aacaacgttg cgcaaactat taactggcga actacttact ctagcttccc 6360
ggcaacaatt aatagactgg atggaggcgg ataaagttgc aggaccactt ctgcgctcgg 6420
cccttccggc tggctggttt attgctgata aatctggagc cggtgagcgt gggtctcgcg 6480
gtatcattgc agcactgggg ccagatggta agccctcccg tatcgtagtt atctacacga 6540
cggggagtca ggcaactatg gatgaacgaa atagacagat cgctgagata ggtgcctcac 6600
tgattaagca ttggtaactg tcagaccaag tttactcata tatactttag attgatttaa 6660
aacttcattt ttaatttaaa aggatctagg tgaagatcct ttttgataat ctcatgacca 6720
aaatccctta acgtgagttt tcgttccact gagcgtcaga ccccgtagaa aagatcaaag 6780
gatcttcttg agatcctttt tttctgcgcg taatctgctg cttgcaaaca aaaaaaccac 6840
cgctaccagc ggtggtttgt ttgccggatc aagagctacc aactcttttt ccgaaggtaa 6900
ctggcttcag cagagcgcag ataccaaata ctgtccttct agtgtagccg tagttaggcc 6960
accacttcaa gaactctgta gcaccgccta catacctcgc tctgctaatc ctgttaccag 7020
tggctgctgc cagtggcgat aagtcgtgtc ttaccgggtt ggactcaaga cgatagttac 7080
cggataaggc gcagcggtcg ggctgaacgg ggggttcgtg cacacagccc agcttggagc 7140
gaacgaccta caccgaactg agatacctac agcgtgagct atgagaaagc gccacgcttc 7200
ccgaagggag aaaggcggac aggtatccgg taagcggcag ggtcggaaca ggagagcgca 7260
cgagggagct tccaggggga aacgcctggt atctttatag tcctgtcggg tttcgccacc 7320
tctgacttga gcgtcgattt ttgtgatgct cgtcaggggg gcggagccta tggaaaaacg 7380
ccagcaacgc ggccttttta cggttcctgg ccttttgctg gccttttgct cacatgttct 7440
ttcctgcgtt atcccctgat tctgtggata accgtattac cgcctttgag tgagctgata 7500
ccgctcgccg cagccgaacg accgagcgca gcgagtcagt gagcgaggaa gcggaagagc 7560
gcccaatacg caaaccgcct ctccccgcgc gttggccgat tcattaatg 7609
<210>57
<211>7492
<212>ДНК
<213>Искусственная последовательность
<220>
<223>Синтезированная конструкция rAAV PD-1 Exon1_MND-BCMACAR
<400>57
cagctgcgcg ctcgctcgct cactgaggcc gcccgggcaa agcccgggcg tcgggcgacc 60
tttggtcgcc cggcctcagt gagcgagcga gcgcgcagag agggagtggc caactccatc 120
actaggggtt ccttgtagtt aatgattaac ccgccatgct acttatctac gtaaggctgt 180
tgcaggcatc acacggtgga aagatctgga actgtggcca tggtgtgagg ccatccacaa 240
ggtggaagct ttgaggggga gccgattagc catggacagt tgtcattcag tagggtcacc 300
tgtgccccag cgaaggggga tgggccggga aggcagaggc caggcacctg cccccagcag 360
gggcagaggc tgtgggcagc cgggaggctc ccagaggctc cgacagaatg ggagtggggt 420
tgagcccacc cctcactgca gcccaggaac ctgagcccag agggggccac ccaccttccc 480
caggcaggga ggcccggccc ccagggagat gggggggatg ggggaggaga agggcctgcc 540
cccacccggc agcctcagga ggggcagctc gggcgggata tggaaagagg ccacagcagt 600
gagcagagac acagaggagg aaggggccct gagctgggga gacccccacg gggtagggcg 660
tgggggccac gggcccacct cctccccatc tcctctgtct ccctgtctct gtctctctct 720
ccctccccca ccctctcccc agtcctaccc cctcctcacc cctcctcccc cagcactgcc 780
tctgtcactc tcgcccacgt ggatgtggag gaagaggggg cgggagcaag gggcgggcac 840
cctcccttca acctgacctg ggacagtttc ccttccgctc acctccgcct gagcagtgga 900
gaaggcggca ctctggtggg gctgctccaa cgcgtaatga aagaccccac ctgtaggttt 960
ggcaagctag gatcaaggtt aggaacagag agacagcaga atatgggcca aacaggatat 1020
ctgtggtaag cagttcctgc cccggctcag ggccaagaac agttggaaca gcagaatatg 1080
ggccaaacag gatatctgtg gtaagcagtt cctgccccgg ctcagggcca agaacagatg 1140
gtccccagat gcggtcccgc cctcagcagt ttctagagaa ccatcagatg tttccagggt 1200
gccccaagga cctgaaatga ccctgtgcct tatttgaact aaccaatcag ttcgcttctc 1260
gcttctgttc gcgcgcttct gctccccgag ctcaataaaa gagcccacaa cccctcactc 1320
ggcgcgattc acctgacgcg tctacgccac catggcactc cccgtcaccg cccttctctt 1380
gcccctcgcc ctgctgctgc atgctgccag gcccgacatt gtgctcactc agtcacctcc 1440
cagcctggcc atgagcctgg gaaaaagggc caccatctcc tgtagagcca gtgagtccgt 1500
cacaatcttg gggagccatc ttattcactg gtatcagcag aagcccgggc agcctccaac 1560
ccttcttatt cagctcgcgt caaacgtcca gacgggtgta cctgccagat tttctggtag 1620
cgggtcccgc actgatttta cactgaccat agatccagtg gaagaagacg atgtggccgt 1680
gtattattgt ctgcagagca gaacgattcc tcgcacattt ggtgggggta ctaagctgga 1740
gattaaggga agcacgtccg gctcagggaa gccgggctcc ggcgagggaa gcacgaaggg 1800
gcaaattcag ctggtccaga gcggacctga gctgaaaaaa cccggcgaga ctgttaagat 1860
cagttgtaaa gcatctggct ataccttcac cgactacagc ataaattggg tgaaacgggc 1920
ccctggaaag ggcctcaaat ggatgggttg gatcaatacc gaaactaggg agcctgctta 1980
tgcatatgac ttccgcggga gattcgcctt ttcactcgag acatctgcct ctactgctta 2040
cctccaaata aacaacctca agtatgaaga tacagccact tacttttgcg ccctcgacta 2100
tagttacgcc atggactact ggggacaggg aacctccgtt accgtcagtt ccgcggccgc 2160
aaccacaaca cctgctccaa ggccccccac acccgctcca actatagcca gccaaccatt 2220
gagcctcaga cctgaagctt gcaggcccgc agcaggaggc gccgtccata cgcgaggcct 2280
ggacttcgcg tgtgatattt atatttgggc ccctttggcc ggaacatgtg gggtgttgct 2340
tctctccctt gtgatcactc tgtattgtaa gcgcgggaga aagaagctcc tgtacatctt 2400
caagcagcct tttatgcgac ctgtgcaaac cactcaggaa gaagatgggt gttcatgccg 2460
cttccccgag gaggaagaag gagggtgtga actgagggtg aaattttcta gaagcgccga 2520
tgctcccgca tatcagcagg gtcagaatca gctctacaat gaattgaatc tcggcaggcg 2580
agaagagtac gatgttctgg acaagagacg gggcagggat cccgagatgg ggggaaagcc 2640
ccggagaaaa aatcctcagg aggggttgta caatgagctg cagaaggaca agatggctga 2700
agcctatagc gagatcggaa tgaaaggcga aagacgcaga ggcaaggggc atgacggtct 2760
gtaccagggt ctctctacag ccaccaagga cacttatgat gcgttgcata tgcaagcctt 2820
gccaccccgc taagcggccg cgctttattt gtgaaatttg tgatgctatt gctttatttg 2880
taaccattat aagctgcaat aaacaagtta acaacaacaa ttgcattcat tttatgtttc 2940
aggttcaggg ggagatgtgg gaggtttttt aaagctcacc ggttctgggc ggtgctacaa 3000
ctgggctggc ggccaggatg gttcttaggt aggtggggtc ggcggtcagg tgtcccagag 3060
ccaggggtct ggagggacct tccaccctca gtccctggca ggtcgggggg tgctgaggcg 3120
ggcctggccc tggcagccca ggggtcccgg agcgaggggt ctggagggac ctttcactct 3180
cagtccctgg caggtcgggg ggtgctgtgg caggcccagc cttggccccc agctctgccc 3240
cttaccctga gctgtgtggc tttgggcagc tcgaactcct gggttcctct ctgggcccca 3300
actcctcccc tggcccaagt cccctctttg ctcctgggca ggcaggacct ctgtcccctc 3360
tcagccggtc cttggggctg cgtgtttctg tagaatgacg ggtcaggctg gccagaaccc 3420
caaaccttgg ccgtggggag tctgcgtggc ggctctgcct tgcccaggca tccttggtcc 3480
tcactcgagt tttcctaagg atgggatgag ccccatgtgg gactaacctt ggctttacga 3540
cgtcaaagtt tagatgagct ggtgatattt ttctcattat atccaaagtg tacctgttcg 3600
agtgaggaca gttcttctgt ctccaggatc cctcctgggt ggggattgtg cccgcctggg 3660
tctctgccca gattccaggg ctctccccga gccctgttca gaccatccgt gggggaggcc 3720
ttggcctcac tcttacgtag ataagtagca tggcgggtta atcattaact acaaggaacc 3780
cctagtgatg gagttggcca ctccctctct gcgcgctcgc tcgctcactg aggccgggcg 3840
accaaaggtc gcccgacgcc cgggctttgc ccgggcggcc tcagtgagcg agcgagcgcg 3900
ccagctggcg taatagcgaa gaggcccgca ccgatcgccc ttcccaacag ttgcgcagcc 3960
tgaatggcga atggcgattc cgttgcaatg gctggcggta atattgttct ggatattacc 4020
agcaaggccg atagtttgag ttcttctact caggcaagtg atgttattac taatcaaaga 4080
agtattgcga caacggttaa tttgcgtgat ggacagactc ttttactcgg tggcctcact 4140
gattataaaa acacttctca ggattctggc gtaccgttcc tgtctaaaat ccctttaatc 4200
ggcctcctgt ttagctcccg ctctgattct aacgaggaaa gcacgttata cgtgctcgtc 4260
aaagcaacca tagtacgcgc cctgtagcgg cgcattaagc gcggcgggtg tggtggttac 4320
gcgcagcgtg accgctacac ttgccagcgc cctagcgccc gctcctttcg ctttcttccc 4380
ttcctttctc gccacgttcg ccggctttcc ccgtcaagct ctaaatcggg ggctcccttt 4440
agggttccga tttagtgctt tacggcacct cgaccccaaa aaacttgatt agggtgatgg 4500
ttcacgtagt gggccatcgc cctgatagac ggtttttcgc cctttgacgt tggagtccac 4560
gttctttaat agtggactct tgttccaaac tggaacaaca ctcaacccta tctcggtcta 4620
ttcttttgat ttataaggga ttttgccgat ttcggcctat tggttaaaaa atgagctgat 4680
ttaacaaaaa tttaacgcga attttaacaa aatattaacg tttacaattt aaatatttgc 4740
ttatacaatc ttcctgtttt tggggctttt ctgattatca accggggtac atatgattga 4800
catgctagtt ttacgattac cgttcatcga ttctcttgtt tgctccagac tctcaggcaa 4860
tgacctgata gcctttgtag agacctctca aaaatagcta ccctctccgg catgaattta 4920
tcagctagaa cggttgaata tcatattgat ggtgatttga ctgtctccgg cctttctcac 4980
ccgtttgaat ctttacctac acattactca ggcattgcat ttaaaatata tgagggttct 5040
aaaaattttt atccttgcgt tgaaataaag gcttctcccg caaaagtatt acagggtcat 5100
aatgtttttg gtacaaccga tttagcttta tgctctgagg ctttattgct taattttgct 5160
aattctttgc cttgcctgta tgatttattg gatgttggaa tcgcctgatg cggtattttc 5220
tccttacgca tctgtgcggt atttcacacc gcatatggtg cactctcagt acaatctgct 5280
ctgatgccgc atagttaagc cagccccgac acccgccaac acccgctgac gcgccctgac 5340
gggcttgtct gctcccggca tccgcttaca gacaagctgt gaccgtctcc gggagctgca 5400
tgtgtcagag gttttcaccg tcatcaccga aacgcgcgag acgaaagggc ctcgtgatac 5460
gcctattttt ataggttaat gtcatgataa taatggtttc ttagacgtca ggtggcactt 5520
ttcggggaaa tgtgcgcgga acccctattt gtttattttt ctaaatacat tcaaatatgt 5580
atccgctcat gagacaataa ccctgataaa tgcttcaata atattgaaaa aggaagagta 5640
tgagtattca acatttccgt gtcgccctta ttcccttttt tgcggcattt tgccttcctg 5700
tttttgctca cccagaaacg ctggtgaaag taaaagatgc tgaagatcag ttgggtgcac 5760
gagtgggtta catcgaactg gatctcaaca gcggtaagat ccttgagagt tttcgccccg 5820
aagaacgttt tccaatgatg agcactttta aagttctgct atgtggcgcg gtattatccc 5880
gtattgacgc cgggcaagag caactcggtc gccgcataca ctattctcag aatgacttgg 5940
ttgagtactc accagtcaca gaaaagcatc ttacggatgg catgacagta agagaattat 6000
gcagtgctgc cataaccatg agtgataaca ctgcggccaa cttacttctg acaacgatcg 6060
gaggaccgaa ggagctaacc gcttttttgc acaacatggg ggatcatgta actcgccttg 6120
atcgttggga accggagctg aatgaagcca taccaaacga cgagcgtgac accacgatgc 6180
ctgtagcaat ggcaacaacg ttgcgcaaac tattaactgg cgaactactt actctagctt 6240
cccggcaaca attaatagac tggatggagg cggataaagt tgcaggacca cttctgcgct 6300
cggcccttcc ggctggctgg tttattgctg ataaatctgg agccggtgag cgtgggtctc 6360
gcggtatcat tgcagcactg gggccagatg gtaagccctc ccgtatcgta gttatctaca 6420
cgacggggag tcaggcaact atggatgaac gaaatagaca gatcgctgag ataggtgcct 6480
cactgattaa gcattggtaa ctgtcagacc aagtttactc atatatactt tagattgatt 6540
taaaacttca tttttaattt aaaaggatct aggtgaagat cctttttgat aatctcatga 6600
ccaaaatccc ttaacgtgag ttttcgttcc actgagcgtc agaccccgta gaaaagatca 6660
aaggatcttc ttgagatcct ttttttctgc gcgtaatctg ctgcttgcaa acaaaaaaac 6720
caccgctacc agcggtggtt tgtttgccgg atcaagagct accaactctt tttccgaagg 6780
taactggctt cagcagagcg cagataccaa atactgtcct tctagtgtag ccgtagttag 6840
gccaccactt caagaactct gtagcaccgc ctacatacct cgctctgcta atcctgttac 6900
cagtggctgc tgccagtggc gataagtcgt gtcttaccgg gttggactca agacgatagt 6960
taccggataa ggcgcagcgg tcgggctgaa cggggggttc gtgcacacag cccagcttgg 7020
agcgaacgac ctacaccgaa ctgagatacc tacagcgtga gctatgagaa agcgccacgc 7080
ttcccgaagg gagaaaggcg gacaggtatc cggtaagcgg cagggtcgga acaggagagc 7140
gcacgaggga gcttccaggg ggaaacgcct ggtatcttta tagtcctgtc gggtttcgcc 7200
acctctgact tgagcgtcga tttttgtgat gctcgtcagg ggggcggagc ctatggaaaa 7260
acgccagcaa cgcggccttt ttacggttcc tggccttttg ctggcctttt gctcacatgt 7320
tctttcctgc gttatcccct gattctgtgg ataaccgtat taccgccttt gagtgagctg 7380
ataccgctcg ccgcagccga acgaccgagc gcagcgagtc agtgagcgag gaagcggaag 7440
agcgcccaat acgcaaaccg cctctccccg cgcgttggcc gattcattaa tg 7492
<210>58
<211>6147
<212>ДНК
<213>Искусственная последовательность
<220>
<223>Синтезированная конструкция rAAV PD-1 Exon1_ATG-mCherry
<400>58
cagctgcgcg ctcgctcgct cactgaggcc gcccgggcaa agcccgggcg tcgggcgacc 60
tttggtcgcc cggcctcagt gagcgagcga gcgcgcagag agggagtggc caactccatc 120
actaggggtt ccttgtagtt aatgattaac ccgccatgct acttatctac gtaaggctgt 180
tgcaggcatc acacggtgga aagatctgga actgtggcca tggtgtgagg ccatccacaa 240
ggtggaagct ttgaggggga gccgattagc catggacagt tgtcattcag tagggtcacc 300
tgtgccccag cgaaggggga tgggccggga aggcagaggc caggcacctg cccccagcag 360
gggcagaggc tgtgggcagc cgggaggctc ccagaggctc cgacagaatg ggagtggggt 420
tgagcccacc cctcactgca gcccaggaac ctgagcccag agggggccac ccaccttccc 480
caggcaggga ggcccggccc ccagggagat gggggggatg ggggaggaga agggcctgcc 540
cccacccggc agcctcagga ggggcagctc gggcgggata tggaaagagg ccacagcagt 600
gagcagagac acagaggagg aaggggccct gagctgggga gacccccacg gggtagggcg 660
tgggggccac gggcccacct cctccccatc tcctctgtct ccctgtctct gtctctctct 720
ccctccccca ccctctcccc agtcctaccc cctcctcacc cctcctcccc cagcactgcc 780
tctgtcactc tcgcccacgt ggatgtggag gaagaggggg cgggagcaag gggcgggcac 840
cctcccttca acctgacctg ggacagtttc ccttccgctc acctccgcct gagcagtgga 900
gaaggcggca ctctggtggg gctgctccag gcatgcaggt gagcaagggc gaggaggata 960
actccgccat catcaaggag ttcctgcgct tcaaggtgca catggagggc tccgtgaacg 1020
gccacgagtt cgagatcgag ggcgagggcg agggccgccc ctacgagggc acccagaccg 1080
ccaagctgaa ggtgaccaag ggtggccccc tgcccttcgc ctgggacatc ctgtcccctc 1140
agttcatgta cggctccaag gcctacgtga agcaccccgc cgacatcccc gactacttga 1200
agctgtcctt ccccgagggc ttcaagtggg agcgcgtgat gaacttcgag gacggcggcg 1260
tggtgaccgt gacccaggac tcctctctgc aggacggcga gttcatctac aaggtgaagc 1320
tgcgcggcac caacttcccc tccgacggcc ccgtaatgca gaagaagacc atgggctggg 1380
aggcctcctc cgagcggatg taccccgagg acggcgccct gaagggcgag atcaagcaga 1440
ggctgaagct gaaggacggc ggccactacg acgctgaggt caagaccacc tacaaggcca 1500
agaagcccgt gcagctgccc ggcgcctaca acgtcaacat caagttggac atcacctccc 1560
acaacgagga ctacaccatc gtggaacagt acgaacgcgc cgagggccgc cactccaccg 1620
gcggcatgga cgagctgtac aagtgatgaa agctttgctt tatttgtgaa atttgtgatg 1680
ctatgcttta tttgtaacca ttataagctg caataaacaa gtttaacaac aacaattgca 1740
ttcattttat gtttcaggtt cagggggagg tgtgggaggt tttttaaagt aggtggggtc 1800
ggcggtcagg tgtcccagag ccaggggtct ggagggacct tccaccctca gtccctggca 1860
ggtcgggggg tgctgaggcg ggcctggccc tggcagccca ggggtcccgg agcgaggggt 1920
ctggagggac ctttcactct cagtccctgg caggtcgggg ggtgctgtgg caggcccagc 1980
cttggccccc agctctgccc cttaccctga gctgtgtggc tttgggcagc tcgaactcct 2040
gggttcctct ctgggcccca actcctcccc tggcccaagt cccctctttg ctcctgggca 2100
ggcaggacct ctgtcccctc tcagccggtc cttggggctg cgtgtttctg tagaatgacg 2160
ggtcaggctg gccagaaccc caaaccttgg ccgtggggag tctgcgtggc ggctctgcct 2220
tgcccaggca tccttggtcc tcactcgagt tttcctaagg atgggatgag ccccatgtgg 2280
gactaacctt ggctttacga cgtcaaagtt tagatgagct ggtgatattt ttctcattat 2340
atccaaagtg tacctgttcg agtgaggaca gttcttctgt ctccaggata cgtagataag 2400
tagcatggcg ggttaatcat taactacaag gaacccctag tgatggagtt ggccactccc 2460
tctctgcgcg ctcgctcgct cactgaggcc gggcgaccaa aggtcgcccg acgcccgggc 2520
tttgcccggg cggcctcagt gagcgagcga gcgcgccagc tggcgtaata gcgaagaggc 2580
ccgcaccgat cgcccttccc aacagttgcg cagcctgaat ggcgaatggc gattccgttg 2640
caatggctgg cggtaatatt gttctggata ttaccagcaa ggccgatagt ttgagttctt 2700
ctactcaggc aagtgatgtt attactaatc aaagaagtat tgcgacaacg gttaatttgc 2760
gtgatggaca gactctttta ctcggtggcc tcactgatta taaaaacact tctcaggatt 2820
ctggcgtacc gttcctgtct aaaatccctt taatcggcct cctgtttagc tcccgctctg 2880
attctaacga ggaaagcacg ttatacgtgc tcgtcaaagc aaccatagta cgcgccctgt 2940
agcggcgcat taagcgcggc gggtgtggtg gttacgcgca gcgtgaccgc tacacttgcc 3000
agcgccctag cgcccgctcc tttcgctttc ttcccttcct ttctcgccac gttcgccggc 3060
tttccccgtc aagctctaaa tcgggggctc cctttagggt tccgatttag tgctttacgg 3120
cacctcgacc ccaaaaaact tgattagggt gatggttcac gtagtgggcc atcgccctga 3180
tagacggttt ttcgcccttt gacgttggag tccacgttct ttaatagtgg actcttgttc 3240
caaactggaa caacactcaa ccctatctcg gtctattctt ttgatttata agggattttg 3300
ccgatttcgg cctattggtt aaaaaatgag ctgatttaac aaaaatttaa cgcgaatttt 3360
aacaaaatat taacgtttac aatttaaata tttgcttata caatcttcct gtttttgggg 3420
cttttctgat tatcaaccgg ggtacatatg attgacatgc tagttttacg attaccgttc 3480
atcgattctc ttgtttgctc cagactctca ggcaatgacc tgatagcctt tgtagagacc 3540
tctcaaaaat agctaccctc tccggcatga atttatcagc tagaacggtt gaatatcata 3600
ttgatggtga tttgactgtc tccggccttt ctcacccgtt tgaatcttta cctacacatt 3660
actcaggcat tgcatttaaa atatatgagg gttctaaaaa tttttatcct tgcgttgaaa 3720
taaaggcttc tcccgcaaaa gtattacagg gtcataatgt ttttggtaca accgatttag 3780
ctttatgctc tgaggcttta ttgcttaatt ttgctaattc tttgccttgc ctgtatgatt 3840
tattggatgt tggaatcgcc tgatgcggta ttttctcctt acgcatctgt gcggtatttc 3900
acaccgcata tggtgcactc tcagtacaat ctgctctgat gccgcatagt taagccagcc 3960
ccgacacccg ccaacacccg ctgacgcgcc ctgacgggct tgtctgctcc cggcatccgc 4020
ttacagacaa gctgtgaccg tctccgggag ctgcatgtgt cagaggtttt caccgtcatc 4080
accgaaacgc gcgagacgaa agggcctcgt gatacgccta tttttatagg ttaatgtcat 4140
gataataatg gtttcttaga cgtcaggtgg cacttttcgg ggaaatgtgc gcggaacccc 4200
tatttgttta tttttctaaa tacattcaaa tatgtatccg ctcatgagac aataaccctg 4260
ataaatgctt caataatatt gaaaaaggaa gagtatgagt attcaacatt tccgtgtcgc 4320
ccttattccc ttttttgcgg cattttgcct tcctgttttt gctcacccag aaacgctggt 4380
gaaagtaaaa gatgctgaag atcagttggg tgcacgagtg ggttacatcg aactggatct 4440
caacagcggt aagatccttg agagttttcg ccccgaagaa cgttttccaa tgatgagcac 4500
ttttaaagtt ctgctatgtg gcgcggtatt atcccgtatt gacgccgggc aagagcaact 4560
cggtcgccgc atacactatt ctcagaatga cttggttgag tactcaccag tcacagaaaa 4620
gcatcttacg gatggcatga cagtaagaga attatgcagt gctgccataa ccatgagtga 4680
taacactgcg gccaacttac ttctgacaac gatcggagga ccgaaggagc taaccgcttt 4740
tttgcacaac atgggggatc atgtaactcg ccttgatcgt tgggaaccgg agctgaatga 4800
agccatacca aacgacgagc gtgacaccac gatgcctgta gcaatggcaa caacgttgcg 4860
caaactatta actggcgaac tacttactct agcttcccgg caacaattaa tagactggat 4920
ggaggcggat aaagttgcag gaccacttct gcgctcggcc cttccggctg gctggtttat 4980
tgctgataaa tctggagccg gtgagcgtgg gtctcgcggt atcattgcag cactggggcc 5040
agatggtaag ccctcccgta tcgtagttat ctacacgacg gggagtcagg caactatgga 5100
tgaacgaaat agacagatcg ctgagatagg tgcctcactg attaagcatt ggtaactgtc 5160
agaccaagtt tactcatata tactttagat tgatttaaaa cttcattttt aatttaaaag 5220
gatctaggtg aagatccttt ttgataatct catgaccaaa atcccttaac gtgagttttc 5280
gttccactga gcgtcagacc ccgtagaaaa gatcaaagga tcttcttgag atcctttttt 5340
tctgcgcgta atctgctgct tgcaaacaaa aaaaccaccg ctaccagcgg tggtttgttt 5400
gccggatcaa gagctaccaa ctctttttcc gaaggtaact ggcttcagca gagcgcagat 5460
accaaatact gtccttctag tgtagccgta gttaggccac cacttcaaga actctgtagc 5520
accgcctaca tacctcgctc tgctaatcct gttaccagtg gctgctgcca gtggcgataa 5580
gtcgtgtctt accgggttgg actcaagacg atagttaccg gataaggcgc agcggtcggg 5640
ctgaacgggg ggttcgtgca cacagcccag cttggagcga acgacctaca ccgaactgag 5700
atacctacag cgtgagctat gagaaagcgc cacgcttccc gaagggagaa aggcggacag 5760
gtatccggta agcggcaggg tcggaacagg agagcgcacg agggagcttc cagggggaaa 5820
cgcctggtat ctttatagtc ctgtcgggtt tcgccacctc tgacttgagc gtcgattttt 5880
gtgatgctcg tcaggggggc ggagcctatg gaaaaacgcc agcaacgcgg cctttttacg 5940
gttcctggcc ttttgctggc cttttgctca catgttcttt cctgcgttat cccctgattc 6000
tgtggataac cgtattaccg cctttgagtg agctgatacc gctcgccgca gccgaacgac 6060
cgagcgcagc gagtcagtga gcgaggaagc ggaagagcgc ccaatacgca aaccgcctct 6120
ccccgcgcgt tggccgattc attaatg 6147
<210>59
<211>232
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Сделано в лаборатории
<400>59
Met Gln Ile Pro Gln Ala Pro Trp Pro Val Val Trp Ala Val Leu Gln
1 5 10 15
Leu Gly Trp Arg Pro Gly Trp Phe Leu Asp Ser Pro Asp Arg Pro Trp
20 25 30
Asn Pro Pro Thr Phe Ser Pro Ala Leu Leu Val Val Thr Glu Gly Asp
35 40 45
Asn Ala Thr Phe Thr Cys Ser Phe Ser Asn Thr Ser Glu Ser Phe Val
50 55 60
Leu Asn Trp Tyr Arg Met Ser Pro Ser Asn Gln Thr Asp Lys Leu Ala
65 70 75 80
Ala Phe Pro Glu Asp Arg Ser Gln Pro Gly Gln Asp Cys Arg Phe Arg
85 90 95
Val Thr Gln Leu Pro Asn Gly Arg Asp Phe His Met Ser Val Val Arg
100 105 110
Ala Arg Arg Asn Asp Ser Gly Thr Tyr Leu Cys Gly Ala Ile Ser Leu
115 120 125
Ala Pro Lys Ala Gln Ile Lys Glu Ser Leu Arg Ala Glu Leu Arg Val
130 135 140
Thr Glu Arg Arg Ala Glu Val Pro Thr Ala His Pro Ser Pro Ser Pro
145 150 155 160
Arg Pro Ala Gly Gln Phe Gln Thr Leu Val Val Gly Val Val Gly Gly
165 170 175
Leu Leu Gly Ser Leu Val Leu Leu Val Trp Val Leu Ala Val Ile Arg
180 185 190
Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr Pro
195 200 205
Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro Pro
210 215 220
Arg Asp Phe Ala Ala Tyr Arg Ser
225 230
<210>60
<211>303
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Синтезированный вариант I-OnuI, PD-1.ile3.exon1_RD2_B1G2
<220>
<221>MOD_RES
<222>(1)..(4)
<223>Любая аминокислота или отсутствует
<220>
<221>MOD_RES
<222>(302)..(303)
<223>Любая аминокислота или отсутствует
<400>60
Xaa Xaa Xaa Xaa Ser Arg Arg Glu Ser Ile Asn Pro Trp Ile Leu Thr
1 5 10 15
Gly Phe Ala Asp Ala Glu Gly Ser Phe Gln Leu Glu Ile Arg Asn Val
20 25 30
Asn Pro Asn Ile Pro Arg Tyr Lys Thr Arg Leu Arg Phe Glu Ile Asp
35 40 45
Leu His Asn Lys Asp Lys Ser Ile Leu Glu Asn Ile Gln Ser Thr Trp
50 55 60
Lys Val Gly Lys Ile Tyr Asn Gln Gly Asp Ser Tyr Val Lys Leu Arg
65 70 75 80
Val Thr Arg Phe Glu Asp Leu Lys Val Ile Ile Asp His Phe Glu Lys
85 90 95
Tyr Pro Leu Ile Thr Gln Lys Leu Gly Asp Tyr Lys Leu Phe Lys Gln
100 105 110
Ala Phe Ser Val Met Glu Asn Lys Glu His Leu Lys Glu Asn Gly Ile
115 120 125
Lys Glu Leu Val Arg Ile Lys Ala Lys Met Asn Trp Gly Leu Asn Asp
130 135 140
Glu Leu Lys Lys Ala Phe Pro Glu Asn Ile Ser Lys Glu Arg Pro Leu
145 150 155 160
Ile Asn Lys Asn Ile Pro Asn Leu Lys Trp Leu Ala Gly Phe Thr Ser
165 170 175
Gly Glu Gly His Phe Gly Val Ile Leu Ala Lys Arg Arg Pro Ala Ser
180 185 190
Pro Val Gln Val Arg Leu Arg Phe Ala Ile Gly Gln His Ile Arg Asp
195 200 205
Lys Asn Leu Met Asn Ser Leu Ile Thr Tyr Leu Gly Cys Gly Arg Ile
210 215 220
Arg Glu Lys Asn Ile Ser Glu Lys Ser Trp Leu Glu Phe Glu Val Thr
225 230 235 240
Lys Phe Ser Asp Ile Asn Asp Lys Ile Ile Pro Val Phe Gln Glu Asn
245 250 255
Thr Leu Ile Gly Val Lys Leu Glu Asp Phe Glu Asp Trp Cys Lys Val
260 265 270
Ala Lys Leu Ile Glu Glu Lys Lys His Leu Thr Glu Ser Gly Leu Asp
275 280 285
Glu Ile Lys Lys Ile Lys Leu Asn Met Asn Lys Gly Arg Xaa Xaa
290 295 300
<210>61
<211>303
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Синтезированный вариант I-OnuI, PD-1.ile3.exon1_RD3_B1G2C4
<220>
<221>MOD_RES
<222>(1)..(4)
<223>Любая аминокислота или отсутствует
<220>
<221>MOD_RES
<222>(302)..(303)
<223>Любая аминокислота или отсутствует
<400>61
Xaa Xaa Xaa Xaa Ser Arg Arg Glu Ser Ile Asn Pro Trp Ile Leu Thr
1 5 10 15
Gly Phe Ala Asp Ala Glu Gly Ser Phe Gly Leu Ser Ile Leu Asn Arg
20 25 30
Asn Arg Gly Thr Ala Arg Tyr His Thr Arg Leu Ser Phe Thr Ile Met
35 40 45
Leu His Asn Lys Asp Lys Ser Ile Leu Glu Asn Ile Gln Ser Thr Trp
50 55 60
Lys Val Gly Ser Ile Leu Asn Asn Gly Asp His Tyr Val Ser Leu Val
65 70 75 80
Val Tyr Arg Phe Glu Asp Leu Lys Val Ile Ile Asp His Phe Glu Lys
85 90 95
Tyr Pro Leu Ile Thr Gln Lys Leu Gly Asp Tyr Lys Leu Phe Lys Gln
100 105 110
Ala Phe Ser Val Met Glu Asn Lys Glu His Leu Lys Glu Asn Gly Ile
115 120 125
Lys Glu Leu Val Arg Ile Lys Ala Lys Met Asn Trp Gly Leu Asn Asp
130 135 140
Glu Leu Lys Lys Ala Phe Pro Glu Asn Ile Ser Lys Glu Arg Pro Leu
145 150 155 160
Ile Asn Lys Asn Ile Pro Asn Phe Lys Trp Leu Ala Gly Phe Thr Ser
165 170 175
Gly Asp Gly Ser Phe Phe Val Arg Leu Arg Lys Ser Asn Val Asn Ala
180 185 190
Arg Val Arg Val Gln Leu Val Phe Glu Ile Ser Gln His Ile Arg Asp
195 200 205
Lys Asn Leu Met Asn Ser Leu Ile Thr Tyr Leu Gly Cys Gly His Ile
210 215 220
Tyr Glu Gly Asn Lys Ser Glu Arg Ser Trp Leu Gln Phe Arg Val Glu
225 230 235 240
Lys Phe Ser Asp Ile Asn Asp Lys Ile Ile Pro Val Phe Gln Glu Asn
245 250 255
Thr Leu Ile Gly Val Lys Leu Glu Asp Phe Glu Asp Trp Cys Lys Val
260 265 270
Ala Lys Leu Ile Glu Glu Lys Lys His Leu Thr Glu Ser Gly Leu Asp
275 280 285
Glu Ile Lys Lys Ile Lys Leu Asn Met Asn Lys Gly Arg Xaa Xaa
290 295 300
<210>62
<211>303
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Синтезированный вариант I-OnuI, PD-1.ile3.exon1_RD3_B1 G2C11
<220>
<221>MOD_RES
<222>(1)..(4)
<223>Любая аминокислота или отсутствует
<220>
<221>MOD_RES
<222>(302)..(303)
<223>Любая аминокислота или отсутствует
<400>62
Xaa Xaa Xaa Xaa Ser Arg Arg Glu Ser Ile Asn Pro Trp Ile Leu Thr
1 5 10 15
Gly Phe Ala Asp Ala Glu Gly Ser Phe Gly Leu Ser Ile Leu Asn Arg
20 25 30
Asn Arg Gly Thr Gly Arg Tyr His Thr Arg Leu Ser Phe Thr Ile Met
35 40 45
Leu His Asn Lys Asp Lys Ser Ile Leu Glu Asn Ile Gln Ser Thr Trp
50 55 60
Lys Val Gly Ser Ile Thr Asn Asn Gly Asp His Tyr Val Ser Leu Val
65 70 75 80
Val Tyr Arg Phe Glu Asp Leu Lys Val Ile Ile Asp His Phe Glu Lys
85 90 95
Tyr Pro Leu Ile Thr Gln Lys Leu Gly Asp Tyr Lys Leu Phe Lys Gln
100 105 110
Ala Phe Ser Val Met Glu Asn Lys Glu His Leu Lys Glu Asn Gly Ile
115 120 125
Lys Glu Leu Val Arg Ile Lys Ala Lys Met Asn Trp Gly Leu Asn Asp
130 135 140
Glu Leu Lys Lys Ala Phe Pro Glu Asn Ile Ser Lys Glu Arg Pro Leu
145 150 155 160
Ile Asn Lys Asn Ile Pro Asn Phe Lys Trp Leu Ala Gly Phe Thr Ser
165 170 175
Gly Asp Gly Ser Phe Phe Val Arg Leu Arg Lys Ser Asn Val Asn Ala
180 185 190
Arg Val Arg Val Gln Leu Val Phe Glu Ile Ser Gln His Ile Arg Asp
195 200 205
Lys Asn Leu Met Asn Ser Leu Ile Thr Tyr Leu Gly Cys Gly His Ile
210 215 220
Tyr Glu Gly Asn Lys Ser Glu Arg Ser Trp Leu Gln Phe Arg Val Glu
225 230 235 240
Lys Phe Ser Asp Ile Asn Asp Lys Ile Ile Pro Val Phe Gln Glu Asn
245 250 255
Thr Leu Ile Gly Val Lys Leu Glu Asp Phe Glu Asp Trp Cys Lys Val
260 265 270
Ala Lys Leu Ile Glu Glu Lys Lys His Leu Thr Glu Ser Gly Leu Asp
275 280 285
Glu Ile Lys Lys Ile Lys Leu Asn Met Asn Lys Gly Arg Xaa Xaa
290 295 300
<210>63
<211>303
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Синтезированный вариант I-OnuI, PD-1.ile3.exon1_RD3_B1 G2C5
<220>
<221>MOD_RES
<222>(1)..(4)
<223>Любая аминокислота или отсутствует
<220>
<221>MOD_RES
<222>(302)..(303)
<223>Любая аминокислота или отсутствует
<400>63
Xaa Xaa Xaa Xaa Ser Arg Arg Glu Ser Ile Asn Pro Trp Ile Leu Thr
1 5 10 15
Gly Phe Ala Asp Ala Glu Gly Ser Phe Gly Leu Ser Ile Leu Asn Arg
20 25 30
Asn Arg Gly Thr Ala Arg Tyr His Thr Arg Leu Ser Phe Thr Ile Met
35 40 45
Leu His Asn Lys Asp Lys Ser Ile Leu Glu Asn Ile Gln Ser Thr Trp
50 55 60
Lys Val Gly Ser Ile Tyr Asn Asn Gly Asp His Tyr Val Ser Leu Glu
65 70 75 80
Val Phe Arg Phe Glu Asp Leu Lys Val Ile Ile Asp His Phe Glu Lys
85 90 95
Tyr Pro Leu Ile Thr Gln Lys Leu Gly Asp Tyr Lys Leu Phe Lys Gln
100 105 110
Ala Phe Ser Val Met Glu Asn Lys Glu His Leu Lys Glu Asn Gly Ile
115 120 125
Lys Glu Leu Val Arg Ile Lys Ala Lys Met Asn Trp Gly Leu Asn Asp
130 135 140
Glu Leu Lys Lys Ala Phe Pro Glu Asn Ile Ser Lys Glu Arg Pro Leu
145 150 155 160
Ile Asn Lys Asn Ile Pro Asn Phe Lys Trp Leu Ala Gly Phe Thr Ser
165 170 175
Gly Asp Gly Ser Phe Phe Val Arg Leu Arg Lys Ser Asn Val Asn Ala
180 185 190
Arg Val Arg Val Gln Leu Val Phe Glu Ile Ser Gln His Ile Arg Asp
195 200 205
Lys Asn Leu Met Asn Ser Leu Ile Thr Tyr Leu Gly Cys Gly His Ile
210 215 220
Tyr Glu Gly Asn Lys Ser Glu Arg Ser Trp Leu Gln Phe Arg Val Glu
225 230 235 240
Lys Phe Ser Asp Ile Asn Asp Lys Ile Ile Pro Val Phe Gln Glu Asn
245 250 255
Thr Leu Ile Gly Val Lys Leu Glu Asp Phe Glu Asp Trp Cys Lys Val
260 265 270
Ala Lys Leu Ile Glu Glu Lys Lys His Leu Thr Glu Ser Gly Leu Asp
275 280 285
Glu Ile Lys Lys Ile Lys Leu Asn Met Asn Lys Gly Arg Xaa Xaa
290 295 300
<210>64
<211>943
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Синтезированная конструкция megaTAL PD-1.ile3.exon1_RD1_B1G2
<400>64
Met Gly Ser Ala Pro Pro Lys Lys Lys Arg Lys Val Val Asp Leu Arg
1 5 10 15
Thr Leu Gly Tyr Ser Gln Gln Gln Gln Glu Lys Ile Lys Pro Lys Val
20 25 30
Arg Ser Thr Val Ala Gln His His Glu Ala Leu Val Gly His Gly Phe
35 40 45
Thr His Ala His Ile Val Ala Leu Ser Gln His Pro Ala Ala Leu Gly
50 55 60
Thr Val Ala Val Thr Tyr Gln His Ile Ile Thr Ala Leu Pro Glu Ala
65 70 75 80
Thr His Glu Asp Ile Val Gly Val Gly Lys Gln Trp Ser Gly Ala Arg
85 90 95
Ala Leu Glu Ala Leu Leu Thr Asp Ala Gly Glu Leu Arg Gly Pro Pro
100 105 110
Leu Gln Leu Asp Thr Gly Gln Leu Val Lys Ile Ala Lys Arg Gly Gly
115 120 125
Val Thr Ala Met Glu Ala Val His Ala Ser Arg Asn Ala Leu Thr Gly
130 135 140
Ala Pro Leu Asn Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn
145 150 155 160
Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
165 170 175
Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala
180 185 190
Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
195 200 205
Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala
210 215 220
Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
225 230 235 240
Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val
245 250 255
Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val
260 265 270
Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp
275 280 285
Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu
290 295 300
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr
305 310 315 320
Pro Asp Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala
325 330 335
Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly
340 345 350
Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys
355 360 365
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Asp
370 375 380
His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser His Asp Gly
385 390 395 400
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
405 410 415
Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala Ser Asn
420 425 430
Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
435 440 445
Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala Ile Ala
450 455 460
Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
465 470 475 480
Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val Val Ala
485 490 495
Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
500 505 510
Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp Gln Val
515 520 525
Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val
530 535 540
Gln Arg Leu Leu Pro Val Leu Cys Gln Asp His Gly Leu Thr Pro Asp
545 550 555 560
Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu
565 570 575
Ser Ile Val Ala Gln Leu Ser Arg Pro Asp Pro Ala Leu Ala Ala Leu
580 585 590
Thr Asn Asp His Leu Val Ala Leu Ala Cys Leu Gly Gly Arg Pro Ala
595 600 605
Met Asp Ala Val Lys Lys Gly Leu Pro His Ala Pro Glu Leu Ile Arg
610 615 620
Arg Val Asn Arg Arg Ile Gly Glu Arg Thr Ser His Arg Val Ala Ile
625 630 635 640
Ser Arg Val Gly Gly Ser Ser Arg Arg Glu Ser Ile Asn Pro Trp Ile
645 650 655
Leu Thr Gly Phe Ala Asp Ala Glu Gly Ser Phe Gly Leu Ser Ile Leu
660 665 670
Asn Arg Asn Arg Gly Thr Ala Arg Tyr His Thr Arg Leu Ser Phe Thr
675 680 685
Ile Met Leu His Asn Lys Asp Lys Ser Ile Leu Glu Asn Ile Gln Ser
690 695 700
Thr Trp Lys Val Gly Ile Ile Thr Asn Asn Gly Asp His Tyr Val Thr
705 710 715 720
Leu Arg Val Thr Arg Phe Glu Asp Leu Lys Val Ile Ile Asp His Phe
725 730 735
Glu Lys Tyr Pro Leu Val Thr Gln Lys Leu Gly Asp Tyr Lys Leu Phe
740 745 750
Lys Gln Ala Phe Ser Val Met Glu Asn Lys Glu His Leu Lys Glu Asn
755 760 765
Gly Ile Lys Glu Leu Val Arg Ile Lys Ala Lys Met Asn Trp Gly Leu
770 775 780
Asn Asp Glu Leu Lys Lys Ala Phe Pro Glu Asn Ile Ser Lys Glu Arg
785 790 795 800
Pro Leu Ile Asn Lys Asn Ile Pro Asn Phe Lys Trp Leu Ala Gly Phe
805 810 815
Thr Ser Gly Asp Gly Ser Phe Phe Val Arg Leu Arg Lys Ser Asn Val
820 825 830
Asn Ala Arg Val Arg Val Gln Leu Val Phe Glu Ile Ser Gln His Ile
835 840 845
Arg Asp Lys Asn Leu Met Asn Ser Leu Ile Thr Tyr Leu Gly Cys Gly
850 855 860
His Ile Tyr Glu Gly Asn Lys Ser Glu Arg Ser Trp Leu Gln Phe Arg
865 870 875 880
Val Glu Lys Phe Ser Asp Ile Asn Asp Lys Ile Ile Pro Val Phe Gln
885 890 895
Glu Asn Thr Leu Ile Gly Val Lys Leu Glu Asp Phe Glu Asp Trp Cys
900 905 910
Lys Val Ala Lys Leu Ile Glu Glu Lys Lys His Leu Thr Glu Ser Gly
915 920 925
Leu Asp Glu Ile Lys Lys Ile Lys Leu Asn Met Asn Lys Gly Arg
930 935 940
<210>65
<211>2832
<212>РНК
<213>Искусственная последовательность
<220>
<223>Синтезированная конструкция мРНК megaTAL PD-1 Exon1 RD2_B1G2
<400>65
augggaagcg cgccaccuaa gaagaaacgc aaagucgugg aucuacgcac gcucggcuac 60
agucagcagc agcaagagaa gaucaaaccg aaggugcguu cgacaguggc gcagcaccac 120
gaggcacugg ugggccaugg guuuacacac gcgcacaucg uugcgcucag ccaacacccg 180
gcagcguuag ggaccgucgc ugucacguau cagcacauaa ucacggcguu gccagaggcg 240
acacacgaag acaucguugg cgucggcaaa cagugguccg gcgcacgcgc ccuggaggcc 300
uugcucacgg augcggggga guugagaggu ccgccguuac aguuggacac aggccaacuu 360
gugaagauug caaaacgugg cggcgugacc gcaauggagg cagugcaugc aucgcgcaau 420
gcacugacgg gugccccccu gaaccuaacc ccugaucagg uagucgcuau agcuucaaac 480
aacgggggca agcaagcacu ggagaccguu caacgacucc ugccagugcu cugccaagac 540
cacggacuua cgccagauca ggugguugcu auugccucca acaauggcgg gaaacaagcg 600
uuggaaacug ugcagagacu guuaccuguc uugugucaag accacggccu cacgccagau 660
caggugguag ccauagcguc gaauggaggu gguaagcaag cccuugaaac gguccagcgu 720
cuucugccgg uguugugcca ggaccacgga cuaacgccgg aucaggucgu agccauugcu 780
ucaaauaacg gcggcaaaca ggcgcuagag acaguccagc gccucuugcc uguguuaugc 840
caggaucacg gcuuaacccc agaccaaguu guggcuauug caucuaacaa ugguggcaaa 900
caagccuugg agacagugca acgauuacug ccugucuuau gucaggauca uggccugacg 960
cccgaucagg uaguggcaau cgcaucuaau aauggaggua agcaagcacu ggagacuguc 1020
cagagauugu uacccguacu augucaagau caugguuuga cgccugauca gguuguugcg 1080
auagccagca acaacggagg gaaacaggcu cuugaaaccg uacagcgacu ucucccaguc 1140
uugugccaag aucacgggcu uacuccugau caagucguag cuaucgccag ccacgacggu 1200
gggaaacagg cccuggaaac cguacaacgu cuccucccag uacuuuguca agaccacggg 1260
uugacuccgg aucaagucgu cgcgaucgcg agcaauggag gggggaagca ggcgcuggaa 1320
acuguucaga gacugcugcc uguacuuugu caggaccaug gucugacacc ugaccaaguu 1380
guggcgauag ccaguaacaa ugggggaaaa caggcacuag agacgguuca aagguuguug 1440
cccguucugu gccaggacca cggcuugaca ccggaucagg ugguagcuau cgcuucacac 1500
gauggcggaa aacaggcuuu agaaacaguc caaagacuuc ucccaguccu uugucaggac 1560
cacggauuga cuccagauca agucguugcu auugcaagua augguggugg uaagcaagcu 1620
uuagaaaccg uacagaggcu uuugccagug cugugccagg accauggacu gaccccugau 1680
caagugguag caauugcauc ucaugaugga ggaaaacaag cucuggaaag cauuguggcc 1740
cagcugagcc ggccugaucc ggcguuggcc gcguugacca acgaccaccu cgucgccuug 1800
gccugccucg gcggacgucc ugccauggau gcagugaaaa agggauugcc gcacgcgccg 1860
gaauugauca gaagagucaa ucgccguauu ggcgaacgca cgucccaucg cguugcgaua 1920
ucuagagugg gaggaagcuc ucgcagagag uccaucaacc cauggauucu gacugguuuc 1980
gcugaugccg aaggaucauu cgggcuaagc auccucaaca gaaacagagg uacugcuaga 2040
uaccacacuc gacugucauu cacaaucaug cugcacaaca aggacaaauc gauucuggag 2100
aauauccagu cgacuuggaa ggucggcaua aucaccaaca acggcgacca cuacgucacc 2160
cugcgcguca cccguuucga agauuugaaa gugauuaucg accacuucga gaaauauccg 2220
cugguaacac agaaauuggg cgauuacaag uuguuuaaac aggcauucag cgucauggag 2280
aacaaagaac aucuuaagga gaaugggauu aaggagcucg uacgaaucaa agcuaagaug 2340
aauugggguc ucaaugacga auugaaaaaa gcauuuccag agaacauuag caaagagcgc 2400
ccccuuauca auaagaacau uccgaauuuc aaauggcugg cuggauucac aucuggugau 2460
ggcuccuucu ucgugcgccu aagaaagucu aauguuaaug cuagaguacg ugugcaacug 2520
guauucgaga ucucacagca caucagagac aagaaccuga ugaauucauu gauaacauac 2580
cuaggcugug gucacaucua cgagggaaac aaaucugagc gcaguuggcu ccaauucaga 2640
guagaaaaau ucagcgauau caacgacaag aucauuccgg uauuccagga aaauacucug 2700
auuggcguca aacucgagga cuuugaagau uggugcaagg uugccaaauu gaucgaagag 2760
aagaaacacc ugaccgaauc cgguuuggau gagauuaaga aaaucaagcu gaacaugaac 2820
aaaggucguu ga 2832
<210>66
<211>2832
<212>РНК
<213>Искусственная последовательность
<220>
<223>Синтезированная конструкция мРНК PD-1 Exon1 RD2_B1G2C4 megaTAL
<400>66
augggaagcg cgccaccuaa gaagaaacgc aaagucgugg aucuacgcac gcucggcuac 60
agucagcagc agcaagagaa gaucaaaccg aaggugcguu cgacaguggc gcagcaccac 120
gaggcacugg ugggccaugg guuuacacac gcgcacaucg uugcgcucag ccaacacccg 180
gcagcguuag ggaccgucgc ugucacguau cagcacauaa ucacggcguu gccagaggcg 240
acacacgaag acaucguugg cgucggcaaa cagugguccg gcgcacgcgc ccuggaggcc 300
uugcucacgg augcggggga guugagaggu ccgccguuac aguuggacac aggccaacuu 360
gugaagauug caaaacgugg cggcgugacc gcaauggagg cagugcaugc aucgcgcaau 420
gcacugacgg gugccccccu gaaccuaacc ccugaucagg uagucgcuau agcuucaaac 480
aacgggggca agcaagcacu ggagaccguu caacgacucc ugccagugcu cugccaagac 540
cacggacuua cgccagauca ggugguugcu auugccucca acaauggcgg gaaacaagcg 600
uuggaaacug ugcagagacu guuaccuguc uugugucaag accacggccu cacgccagau 660
caggugguag ccauagcguc gaauggaggu gguaagcaag cccuugaaac gguccagcgu 720
cuucugccgg uguugugcca ggaccacgga cuaacgccgg aucaggucgu agccauugcu 780
ucaaauaacg gcggcaaaca ggcgcuagag acaguccagc gccucuugcc uguguuaugc 840
caggaucacg gcuuaacccc agaccaaguu guggcuauug caucuaacaa ugguggcaaa 900
caagccuugg agacagugca acgauuacug ccugucuuau gucaggauca uggccugacg 960
cccgaucagg uaguggcaau cgcaucuaau aauggaggua agcaagcacu ggagacuguc 1020
cagagauugu uacccguacu augucaagau caugguuuga cgccugauca gguuguugcg 1080
auagccagca acaacggagg gaaacaggcu cuugaaaccg uacagcgacu ucucccaguc 1140
uugugccaag aucacgggcu uacuccugau caagucguag cuaucgccag ccacgacggu 1200
gggaaacagg cccuggaaac cguacaacgu cuccucccag uacuuuguca agaccacggg 1260
uugacuccgg aucaagucgu cgcgaucgcg agcaauggag gggggaagca ggcgcuggaa 1320
acuguucaga gacugcugcc uguacuuugu caggaccaug gucugacacc ugaccaaguu 1380
guggcgauag ccaguaacaa ugggggaaaa caggcacuag agacgguuca aagguuguug 1440
cccguucugu gccaggacca cggcuugaca ccggaucagg ugguagcuau cgcuucacac 1500
gauggcggaa aacaggcuuu agaaacaguc caaagacuuc ucccaguccu uugucaggac 1560
cacggauuga cuccagauca agucguugcu auugcaagua augguggugg uaagcaagcu 1620
uuagaaaccg uacagaggcu uuugccagug cugugccagg accauggacu gaccccugau 1680
caagugguag caauugcauc ucaugaugga ggaaaacaag cucuggaaag cauuguggcc 1740
cagcugagcc ggccugaucc ggcguuggcc gcguugacca acgaccaccu cgucgccuug 1800
gccugccucg gcggacgucc ugccauggau gcagugaaaa agggauugcc gcacgcgccg 1860
gaauugauca gaagagucaa ucgccguauu ggcgaacgca cgucccaucg cguugcgaua 1920
ucuagagugg gaggaagcuc ucgcagagag uccaucaacc cauggauucu gacugguuuc 1980
gcugaugccg aaggaucauu cgggcuaagc auccucaaca gaaacagagg uacugcuaga 2040
uaccacacuc gacugucauu cacaaucaug cugcacaaca aggacaaauc gauucuggag 2100
aauauccagu cgacuuggaa ggucggcagc auccucaaca auggcgacca cuacgucucg 2160
cugguggucu accguuucga agauuugaaa gugauuaucg accacuucga gaaauauccg 2220
cugauaacac agaaauuggg cgauuacaag uuguuuaaac aggcauucag cgucauggag 2280
aacaaagaac aucuuaagga gaaugggauu aaggagcucg uacgaaucaa agcuaagaug 2340
aauugggguc ucaaugacga auugaaaaaa gcauuuccag agaacauuag caaagagcgc 2400
ccccuuauca auaagaacau uccgaauuuc aaauggcugg cuggauucac aucuggugau 2460
ggcuccuucu ucgugcgccu aagaaagucu aauguuaaug cuagaguacg ugugcaacug 2520
guauucgaga ucucacagca caucagagac aagaaccuga ugaauucauu gauaacauac 2580
cuaggcugug gucacaucua cgagggaaac aaaucugagc gcaguuggcu ccaauucaga 2640
guagaaaaau ucagcgauau caacgacaag aucauuccgg uauuccagga aaauacucug 2700
auuggcguca aacucgagga cuuugaagau uggugcaagg uugccaaauu gaucgaagag 2760
aagaaacacc ugaccgaauc cgguuuggau gagauuaaga aaaucaagcu gaacaugaac 2820
aaaggucguu ga 2832
<210>67
<211>2832
<212>РНК
<213>Искусственная последовательность
<220>
<223>Синтезированная конструкция мРНК megaTAL PD-1 Exon1 RD2_B1G2C11
<400>67
augggaagcg cgccaccuaa gaagaaacgc aaagucgugg aucuacgcac gcucggcuac 60
agucagcagc agcaagagaa gaucaaaccg aaggugcguu cgacaguggc gcagcaccac 120
gaggcacugg ugggccaugg guuuacacac gcgcacaucg uugcgcucag ccaacacccg 180
gcagcguuag ggaccgucgc ugucacguau cagcacauaa ucacggcguu gccagaggcg 240
acacacgaag acaucguugg cgucggcaaa cagugguccg gcgcacgcgc ccuggaggcc 300
uugcucacgg augcggggga guugagaggu ccgccguuac aguuggacac aggccaacuu 360
gugaagauug caaaacgugg cggcgugacc gcaauggagg cagugcaugc aucgcgcaau 420
gcacugacgg gugccccccu gaaccuaacc ccugaucagg uagucgcuau agcuucaaac 480
aacgggggca agcaagcacu ggagaccguu caacgacucc ugccagugcu cugccaagac 540
cacggacuua cgccagauca ggugguugcu auugccucca acaauggcgg gaaacaagcg 600
uuggaaacug ugcagagacu guuaccuguc uugugucaag accacggccu cacgccagau 660
caggugguag ccauagcguc gaauggaggu gguaagcaag cccuugaaac gguccagcgu 720
cuucugccgg uguugugcca ggaccacgga cuaacgccgg aucaggucgu agccauugcu 780
ucaaauaacg gcggcaaaca ggcgcuagag acaguccagc gccucuugcc uguguuaugc 840
caggaucacg gcuuaacccc agaccaaguu guggcuauug caucuaacaa ugguggcaaa 900
caagccuugg agacagugca acgauuacug ccugucuuau gucaggauca uggccugacg 960
cccgaucagg uaguggcaau cgcaucuaau aauggaggua agcaagcacu ggagacuguc 1020
cagagauugu uacccguacu augucaagau caugguuuga cgccugauca gguuguugcg 1080
auagccagca acaacggagg gaaacaggcu cuugaaaccg uacagcgacu ucucccaguc 1140
uugugccaag aucacgggcu uacuccugau caagucguag cuaucgccag ccacgacggu 1200
gggaaacagg cccuggaaac cguacaacgu cuccucccag uacuuuguca agaccacggg 1260
uugacuccgg aucaagucgu cgcgaucgcg agcaauggag gggggaagca ggcgcuggaa 1320
acuguucaga gacugcugcc uguacuuugu caggaccaug gucugacacc ugaccaaguu 1380
guggcgauag ccaguaacaa ugggggaaaa caggcacuag agacgguuca aagguuguug 1440
cccguucugu gccaggacca cggcuugaca ccggaucagg ugguagcuau cgcuucacac 1500
gauggcggaa aacaggcuuu agaaacaguc caaagacuuc ucccaguccu uugucaggac 1560
cacggauuga cuccagauca agucguugcu auugcaagua augguggugg uaagcaagcu 1620
uuagaaaccg uacagaggcu uuugccagug cugugccagg accauggacu gaccccugau 1680
caagugguag caauugcauc ucaugaugga ggaaaacaag cucuggaaag cauuguggcc 1740
cagcugagcc ggccugaucc ggcguuggcc gcguugacca acgaccaccu cgucgccuug 1800
gccugccucg gcggacgucc ugccauggau gcagugaaaa agggauugcc gcacgcgccg 1860
gaauugauca gaagagucaa ucgccguauu ggcgaacgca cgucccaucg cguugcgaua 1920
ucuagagugg gaggaagcuc ucgcagagag uccaucaacc cauggauucu gacugguuuc 1980
gcugaugccg aaggaucauu cgggcuaagc auccucaaca gaaacagagg uacugguaga 2040
uaccacacuc gacugucauu cacaaucaug cugcacaaca aggacaaauc gauucuggag 2100
aauauccagu cgacuuggaa ggucggcucg aucacgaaca acggcgacca cuacgucagc 2160
cuggucgucu accguuucga agauuugaaa gugauuaucg accacuucga gaaauauccg 2220
cugauaacac agaaauuggg cgauuacaag uuguuuaaac aggcauucag cgucauggag 2280
aacaaagaac aucuuaagga gaaugggauu aaggagcucg uacgaaucaa agcuaagaug 2340
aauugggguc ucaaugacga auugaaaaaa gcauuuccag agaacauuag caaagagcgc 2400
ccccuuauca auaagaacau uccgaauuuc aaauggcugg cuggauucac aucuggugau 2460
ggcuccuucu ucgugcgccu aagaaagucu aauguuaaug cuagaguacg ugugcaacug 2520
guauucgaga ucucacagca caucagagac aagaaccuga ugaauucauu gauaacauac 2580
cuaggcugug gucacaucua cgagggaaac aaaucugagc gcaguuggcu ccaauucaga 2640
guagaaaaau ucagcgauau caacgacaag aucauuccgg uauuccagga aaauacucug 2700
auuggcguca aacucgagga cuuugaagau uggugcaagg uugccaaauu gaucgaagag 2760
aagaaacacc ugaccgaauc cgguuuggau gagauuaaga aaaucaagcu gaacaugaac 2820
aaaggucguu ga 2832
<210>68
<211>2832
<212>РНК
<213>Искусственная последовательность
<220>
<223>Синтезированная конструкция мРНК megaTAL PD-1 Exon1 RD2_B1G2C5
<400>68
augggaagcg cgccaccuaa gaagaaacgc aaagucgugg aucuacgcac gcucggcuac 60
agucagcagc agcaagagaa gaucaaaccg aaggugcguu cgacaguggc gcagcaccac 120
gaggcacugg ugggccaugg guuuacacac gcgcacaucg uugcgcucag ccaacacccg 180
gcagcguuag ggaccgucgc ugucacguau cagcacauaa ucacggcguu gccagaggcg 240
acacacgaag acaucguugg cgucggcaaa cagugguccg gcgcacgcgc ccuggaggcc 300
uugcucacgg augcggggga guugagaggu ccgccguuac aguuggacac aggccaacuu 360
gugaagauug caaaacgugg cggcgugacc gcaauggagg cagugcaugc aucgcgcaau 420
gcacugacgg gugccccccu gaaccuaacc ccugaucagg uagucgcuau agcuucaaac 480
aacgggggca agcaagcacu ggagaccguu caacgacucc ugccagugcu cugccaagac 540
cacggacuua cgccagauca ggugguugcu auugccucca acaauggcgg gaaacaagcg 600
uuggaaacug ugcagagacu guuaccuguc uugugucaag accacggccu cacgccagau 660
caggugguag ccauagcguc gaauggaggu gguaagcaag cccuugaaac gguccagcgu 720
cuucugccgg uguugugcca ggaccacgga cuaacgccgg aucaggucgu agccauugcu 780
ucaaauaacg gcggcaaaca ggcgcuagag acaguccagc gccucuugcc uguguuaugc 840
caggaucacg gcuuaacccc agaccaaguu guggcuauug caucuaacaa ugguggcaaa 900
caagccuugg agacagugca acgauuacug ccugucuuau gucaggauca uggccugacg 960
cccgaucagg uaguggcaau cgcaucuaau aauggaggua agcaagcacu ggagacuguc 1020
cagagauugu uacccguacu augucaagau caugguuuga cgccugauca gguuguugcg 1080
auagccagca acaacggagg gaaacaggcu cuugaaaccg uacagcgacu ucucccaguc 1140
uugugccaag aucacgggcu uacuccugau caagucguag cuaucgccag ccacgacggu 1200
gggaaacagg cccuggaaac cguacaacgu cuccucccag uacuuuguca agaccacggg 1260
uugacuccgg aucaagucgu cgcgaucgcg agcaauggag gggggaagca ggcgcuggaa 1320
acuguucaga gacugcugcc uguacuuugu caggaccaug gucugacacc ugaccaaguu 1380
guggcgauag ccaguaacaa ugggggaaaa caggcacuag agacgguuca aagguuguug 1440
cccguucugu gccaggacca cggcuugaca ccggaucagg ugguagcuau cgcuucacac 1500
gauggcggaa aacaggcuuu agaaacaguc caaagacuuc ucccaguccu uugucaggac 1560
cacggauuga cuccagauca agucguugcu auugcaagua augguggugg uaagcaagcu 1620
uuagaaaccg uacagaggcu uuugccagug cugugccagg accauggacu gaccccugau 1680
caagugguag caauugcauc ucaugaugga ggaaaacaag cucuggaaag cauuguggcc 1740
cagcugagcc ggccugaucc ggcguuggcc gcguugacca acgaccaccu cgucgccuug 1800
gccugccucg gcggacgucc ugccauggau gcagugaaaa agggauugcc gcacgcgccg 1860
gaauugauca gaagagucaa ucgccguauu ggcgaacgca cgucccaucg cguugcgaua 1920
ucuagagugg gaggaagcuc ucgcagagag uccaucaacc cauggauucu gacugguuuc 1980
gcugaugccg aaggaucauu cgggcuaagc auccucaaca gaaacagagg uacugcuaga 2040
uaccacacuc gacugucauu cacaaucaug cugcacaaca aggacaaauc gauucuggag 2100
aauauccagu cgacuuggaa ggucggcucg aucuacaaca acggcgacca cuacgucucg 2160
cuggaggucu uccguuucga agauuugaaa gugauuaucg accacuucga gaaauauccg 2220
cugauaacac agaaauuggg cgauuacaag uuguuuaaac aggcauucag cgucauggag 2280
aacaaagaac aucuuaagga gaaugggauu aaggagcucg uacgaaucaa agcuaagaug 2340
aauugggguc ucaaugacga auugaaaaaa gcauuuccag agaacauuag caaagagcgc 2400
ccccuuauca auaagaacau uccgaauuuc aaauggcugg cuggauucac aucuggugau 2460
ggcuccuucu ucgugcgccu aagaaagucu aauguuaaug cuagaguacg ugugcaacug 2520
guauucgaga ucucacagca caucagagac aagaaccuga ugaauucauu gauaacauac 2580
cuaggcugug gucacaucua cgagggaaac aaaucugagc gcaguuggcu ccaauucaga 2640
guagaaaaau ucagcgauau caacgacaag aucauuccgg uauuccagga aaauacucug 2700
auuggcguca aacucgagga cuuugaagau uggugcaagg uugccaaauu gaucgaagag 2760
aagaaacacc ugaccgaauc cgguuuggau gagauuaaga aaaucaagcu gaacaugaac 2820
aaaggucguu ga 2832
<210>69
<211>3
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Иллюстративная линкерная последовательность
<400>69
Gly Gly Gly
1
<210>70
<211>5
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Иллюстративная линкерная последовательность
<400>70
Asp Gly Gly Gly Ser
1 5
<210>71
<211>5
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Иллюстративная линкерная последовательность
<400>71
Thr Gly Glu Lys Pro
1 5
<210>72
<211>4
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Иллюстративная линкерная последовательность
<400>72
Gly Gly Arg Arg
1
<210>73
<211>5
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Иллюстративная линкерная последовательность
<400>73
Gly Gly Gly Gly Ser
1 5
<210>74
<211>14
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Иллюстративная линкерная последовательность
<400>74
Glu Gly Lys Ser Ser Gly Ser Gly Ser Glu Ser Lys Val Asp
1 5 10
<210>75
<211>18
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Иллюстративная линкерная последовательность
<400>75
Lys Glu Ser Gly Ser Val Ser Ser Glu Gln Leu Ala Gln Phe Arg Ser
1 5 10 15
Leu Asp
<210>76
<211>8
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Иллюстративная линкерная последовательность
<400>76
Gly Gly Arg Arg Gly Gly Gly Ser
1 5
<210>77
<211>9
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Иллюстративная линкерная последовательность
<400>77
Leu Arg Gln Arg Asp Gly Glu Arg Pro
1 5
<210>78
<211>12
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Иллюстративная линкерная последовательность
<400>78
Leu Arg Gln Lys Asp Gly Gly Gly Ser Glu Arg Pro
1 5 10
<210>79
<211>16
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Иллюстративная линкерная последовательность
<400>79
Leu Arg Gln Lys Asp Gly Gly Gly Ser Gly Gly Gly Ser Glu Arg Pro
1 5 10 15
<210>80
<211>7
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Cleavage sequence by TEV protease
<220>
<221>прочий_признак
<222>(2)..(3)
<223>Xaa - любая аминокислота
<220>
<221>прочий_признак
<222>(5)..(5)
<223>Xaa - любая аминокислота
<220>
<221>ПРОЧИЙ_ПРИЗНАК
<222>(7)..(7)
<223>Xaa=Gly или Ser
<400>80
Glu Xaa Xaa Tyr Xaa Gln Xaa
1 5
<210>81
<211>7
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Последовательность сайта расщепления протеазой TEV
<400>81
Glu Asn Leu Tyr Phe Gln Gly
1 5
<210>82
<211>7
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Последовательность сайта расщепления протеазой TEV
<400>82
Glu Asn Leu Tyr Phe Gln Ser
1 5
<210>83
<211>22
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Саморасщепляемый полипептид, содержащий сайт 2А
<400>83
Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val
1 5 10 15
Glu Glu Asn Pro Gly Pro
20
<210>84
<211>19
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Саморасщепляемый полипептид, содержащий сайт 2А
<400>84
Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn
1 5 10 15
Pro Gly Pro
<210>85
<211>14
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Саморасщепляемый полипептид, содержащий сайт 2А
<400>85
Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro
1 5 10
<210>86
<211>21
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Саморасщепляемый полипептид, содержащий сайт 2А
<400>86
Gly Ser Gly Glu Gly Arg Gly Ser Leu Leu Thr Cys Gly Asp Val Glu
1 5 10 15
Glu Asn Pro Gly Pro
20
<210>87
<211>18
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Саморасщепляемый полипептид, содержащий сайт 2А
<400>87
Glu Gly Arg Gly Ser Leu Leu Thr Cys Gly Asp Val Glu Glu Asn Pro
1 5 10 15
Gly Pro
<210>88
<211>13
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Саморасщепляемый полипептид, содержащий сайт 2А
<400>88
Leu Leu Thr Cys Gly Asp Val Glu Glu Asn Pro Gly Pro
1 5 10
<210>89
<211>23
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Саморасщепляемый полипептид, содержащий сайт 2А
<400>89
Gly Ser Gly Gln Cys Thr Asn Tyr Ala Leu Leu Lys Leu Ala Gly Asp
1 5 10 15
Val Glu Ser Asn Pro Gly Pro
20
<210>90
<211>20
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Саморасщепляемый полипептид, содержащий сайт 2А
<400>90
Gln Cys Thr Asn Tyr Ala Leu Leu Lys Leu Ala Gly Asp Val Glu Ser
1 5 10 15
Asn Pro Gly Pro
20
<210>91
<211>14
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Саморасщепляемый полипептид, содержащий сайт 2А
<400>91
Leu Leu Lys Leu Ala Gly Asp Val Glu Ser Asn Pro Gly Pro
1 5 10
<210>92
<211>25
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Саморасщепляемый полипептид, содержащий сайт 2А
<400>92
Gly Ser Gly Val Lys Gln Thr Leu Asn Phe Asp Leu Leu Lys Leu Ala
1 5 10 15
Gly Asp Val Glu Ser Asn Pro Gly Pro
20 25
<210>93
<211>22
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Саморасщепляемый полипептид, содержащий сайт 2А
<400>93
Val Lys Gln Thr Leu Asn Phe Asp Leu Leu Lys Leu Ala Gly Asp Val
1 5 10 15
Glu Ser Asn Pro Gly Pro
20
<210>94
<211>14
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Саморасщепляемый полипептид, содержащий сайт 2А
<400>94
Leu Leu Lys Leu Ala Gly Asp Val Glu Ser Asn Pro Gly Pro
1 5 10
<210>95
<211>19
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Саморасщепляемый полипептид, содержащий сайт 2А
<400>95
Leu Leu Asn Phe Asp Leu Leu Lys Leu Ala Gly Asp Val Glu Ser Asn
1 5 10 15
Pro Gly Pro
<210>96
<211>19
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Саморасщепляемый полипептид, содержащий сайт 2А
<400>96
Thr Leu Asn Phe Asp Leu Leu Lys Leu Ala Gly Asp Val Glu Ser Asn
1 5 10 15
Pro Gly Pro
<210>97
<211>14
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Саморасщепляемый полипептид, содержащий сайт 2А
<400>97
Leu Leu Lys Leu Ala Gly Asp Val Glu Ser Asn Pro Gly Pro
1 5 10
<210>98
<211>17
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Саморасщепляемый полипептид, содержащий сайт 2А
<400>98
Asn Phe Asp Leu Leu Lys Leu Ala Gly Asp Val Glu Ser Asn Pro Gly
1 5 10 15
Pro
<210>99
<211>20
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Саморасщепляемый полипептид, содержащий сайт 2А
<400>99
Gln Leu Leu Asn Phe Asp Leu Leu Lys Leu Ala Gly Asp Val Glu Ser
1 5 10 15
Asn Pro Gly Pro
20
<210>100
<211>24
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Саморасщепляемый полипептид, содержащий сайт 2А
<400>100
Ala Pro Val Lys Gln Thr Leu Asn Phe Asp Leu Leu Lys Leu Ala Gly
1 5 10 15
Asp Val Glu Ser Asn Pro Gly Pro
20
<210>101
<211>40
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Саморасщепляемый полипептид, содержащий сайт 2А
<400>101
Val Thr Glu Leu Leu Tyr Arg Met Lys Arg Ala Glu Thr Tyr Cys Pro
1 5 10 15
Arg Pro Leu Leu Ala Ile His Pro Thr Glu Ala Arg His Lys Gln Lys
20 25 30
Ile Val Ala Pro Val Lys Gln Thr
35 40
<210>102
<211>18
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Саморасщепляемый полипептид, содержащий сайт 2А
<400>102
Leu Asn Phe Asp Leu Leu Lys Leu Ala Gly Asp Val Glu Ser Asn Pro
1 5 10 15
Gly Pro
<210>103
<211>40
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Саморасщепляемый полипептид, содержащий сайт 2А
<400>103
Leu Leu Ala Ile His Pro Thr Glu Ala Arg His Lys Gln Lys Ile Val
1 5 10 15
Ala Pro Val Lys Gln Thr Leu Asn Phe Asp Leu Leu Lys Leu Ala Gly
20 25 30
Asp Val Glu Ser Asn Pro Gly Pro
35 40
<210>104
<211>33
<212>БЕЛОК
<213>Искусственная последовательность
<220>
<223>Саморасщепляемый полипептид, содержащий сайт 2А
<400>104
Glu Ala Arg His Lys Gln Lys Ile Val Ala Pro Val Lys Gln Thr Leu
1 5 10 15
Asn Phe Asp Leu Leu Lys Leu Ala Gly Asp Val Glu Ser Asn Pro Gly
20 25 30
Pro
<210>105
<211>10
<212>ДНК
<213>Искусственная последовательность
<220>
<223>Консенсусная последовательность Козака
<400>105
gccrccatgg 10
<210>106
<211>8
<212>БЕЛОК
<213>Homo sapiens
<400>106
Glu Gln Thr Glu Tyr Ala Thr Ile
1 5
<210>107
<211>43
<212>ДНК
<213>Homo sapiens
<400>107
tgagcagacg gagtatgcca ccattgtctt tcctagcgga atg 43
<210>108
<211>43
<212>ДНК
<213>Искусственная последовательность
<220>
<223>Сделано в лаборатории
<400>108
cattccgcta ggaaagacaa tggtggcata ctccgtctgc tca 43
<210>109
<211>22
<212>ДНК
<213>Искусственная последовательность
<220>
<223>Сделано в лаборатории
<220>
<221>прочий_признак
<222>(14)..(22)
<223>n is A, C, G или T
<400>109
aatggtggca tacnnnnnnn nn 22
<210>110
<211>22
<212>ДНК
<213>Искусственная последовательность
<220>
<223>Сделано в лаборатории
<220>
<221>прочий_признак
<222>(1)..(9)
<223>n is a, c, g, or t
<400>110
nnnnnnnnna tactccgtct gc 22
<210>111
<211>22
<212>ДНК
<213>Искусственная последовательность
<220>
<223>Сделано в лаборатории
<400>111
aatggtggca tactccgtct gc 22
<210>112
<211>43
<212>ДНК
<213>Homo sapiens
<400>112
attccgctag gaaagacaat ggtggcatac tccgtctgct cag 43
<210>113
<211>43
<212>ДНК
<213>Искусственная последовательность
<220>
<223>Сделано в лаборатории
<400>113
atttcgttag gaaagataat ggtggtatat ttcgtttgtt tag 43
<210>114
<211>7
<212>БЕЛОК
<213>Homo sapiens
<400>114
Met Gln Ile Pro Gln Ala Pro
1 5
<210>115
<211>44
<212>ДНК
<213>Homo sapiens
<400>115
ctctggtggg gctgctccag gcatgcagat cccacaggcg ccct 44
<210>116
<211>44
<212>ДНК
<213>Искусственная последовательность
<220>
<223>Сделано в лаборатории
<400>116
agggcgcctg tgggatctgc atccctggag cagccccacc agag 44
<210>117
<211>8
<212>БЕЛОК
<213>Homo sapiens
<400>117
Asn Gly Arg Asp Phe His Met Ser
1 5
<210>118
<211>43
<212>ДНК
<213>Homo sapiens
<400>118
gtgtcacaca actgcccaac gggcgtgact tccacatgag cgt 43
<210>119
<211>43
<212>ДНК
<213>Искусственная последовательность
<220>
<223>Сделано в лаборатории
<400>119
acgctcatgt ggaagtcacg cccgttgggc agttgtgtga cac 43
<210>120
<211>32
<212>ДНК
<213>Homo sapiens
<400>120
gctccaggca tgcagatccc acaggcgccc tg 32
<210>121
<211>32
<212>ДНК
<213>Искусственная последовательность
<220>
<223>Сделано в лаборатории
<400>121
actccaaaca tacaaatccc acaaacaccc ta 32
<210>122
<211>31
<212>ДНК
<213>Homo sapiens
<400>122
gcccaacggg cgtgacttcc acatgagcgt g 31
<210>123
<211>31
<212>ДНК
<213>Искусственная последовательность
<220>
<223>Сделано в лаборатории
<400>123
acccaacgaa cgtaacttcc acataaacgt a 31
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ВАРИАНТЫ, КОМПОЗИЦИИ И СПОСОБЫ ПРИМЕНЕНИЯ ЭНДОНУКЛЕАЗЫ CBLB | 2018 |
|
RU2779097C2 |
| ПОЛИФУНКЦИОНАЛЬНЫЕ БЕЛКОВЫЕ МОЛЕКУЛЫ, СОДЕРЖАЩИЕ ДЕКОРИН, И ИХ ПРИМЕНЕНИЕ | 2019 |
|
RU2823357C2 |
| Анти-PD-L1 и IL-2 цитокины | 2017 |
|
RU2769282C2 |
| PD-L1 специфические антитела | 2017 |
|
RU2756236C2 |
| ДОСТАВКА ТЕРАПЕВТИЧЕСКИХ ПОЛИПЕПТИДОВ ПОСРЕДСТВОМ ПСЕВДОТИПИРОВАННЫХ ОНКОЛИТИЧЕСКИХ ВИРУСОВ | 2017 |
|
RU2758007C2 |
| МНОГОФУНКЦИОНАЛЬНЫЕ СЛИТЫЕ БЕЛКИ И ИХ ПРИМЕНЕНИЯ | 2020 |
|
RU2815388C2 |
| АНТИ-PD-L1 АНТИТЕЛО ДЛЯ ДЕТЕКЦИИ PD-L1 | 2018 |
|
RU2758723C2 |
| СВЯЗЫВАЮЩИЕСЯ С PD-1 АНТИТЕЛА И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2019 |
|
RU2812280C1 |
| СЛИТЫЙ БЕЛОК И ЕГО ПРИМЕНЕНИЕ | 2020 |
|
RU2811120C2 |
| АНТИТЕЛА, БЛОКИРУЮЩИЕ CD73 | 2020 |
|
RU2819204C2 |
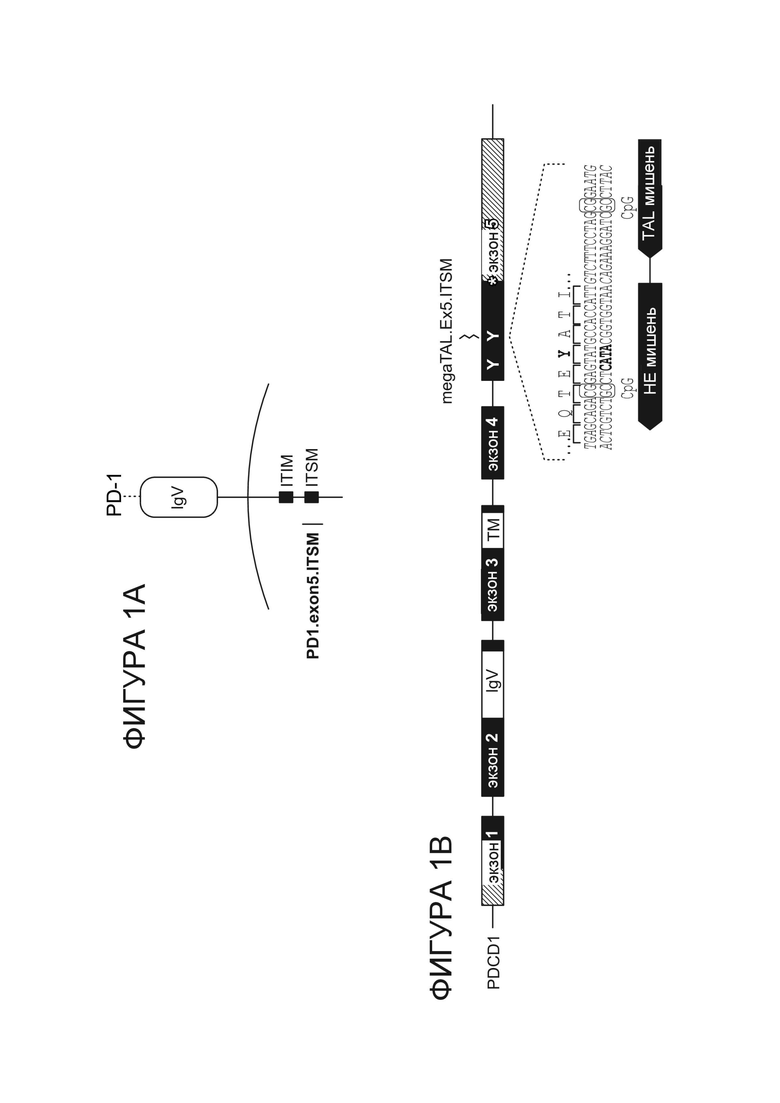

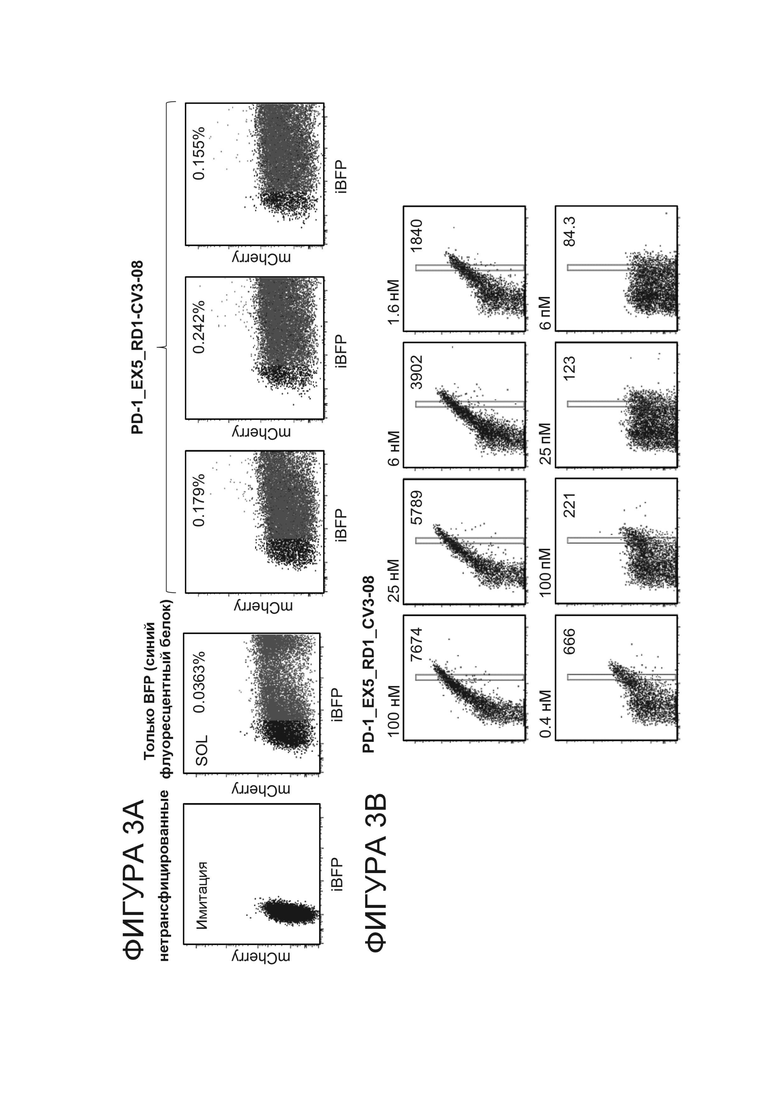

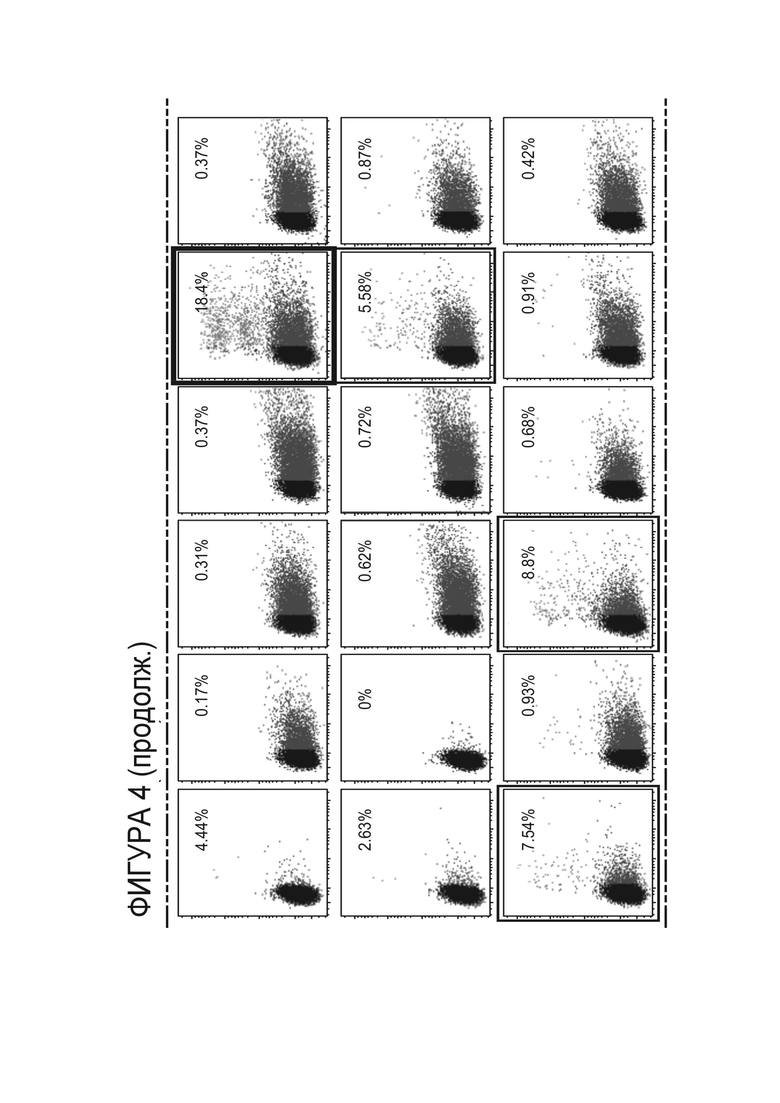
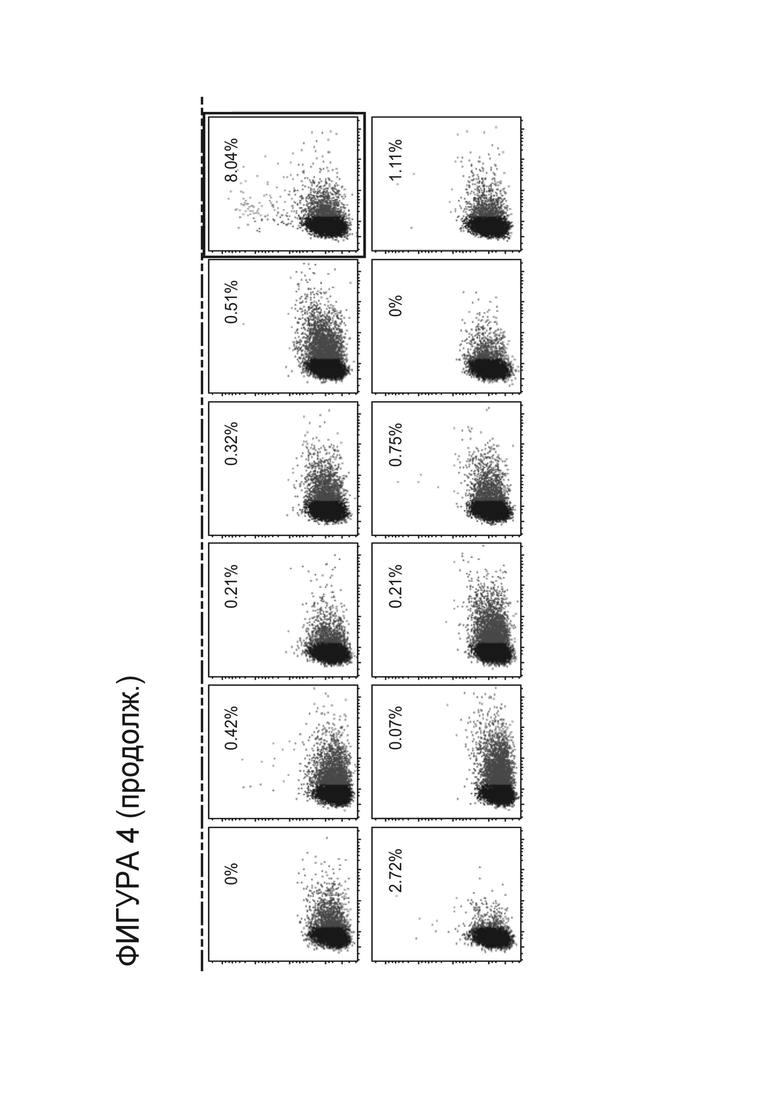

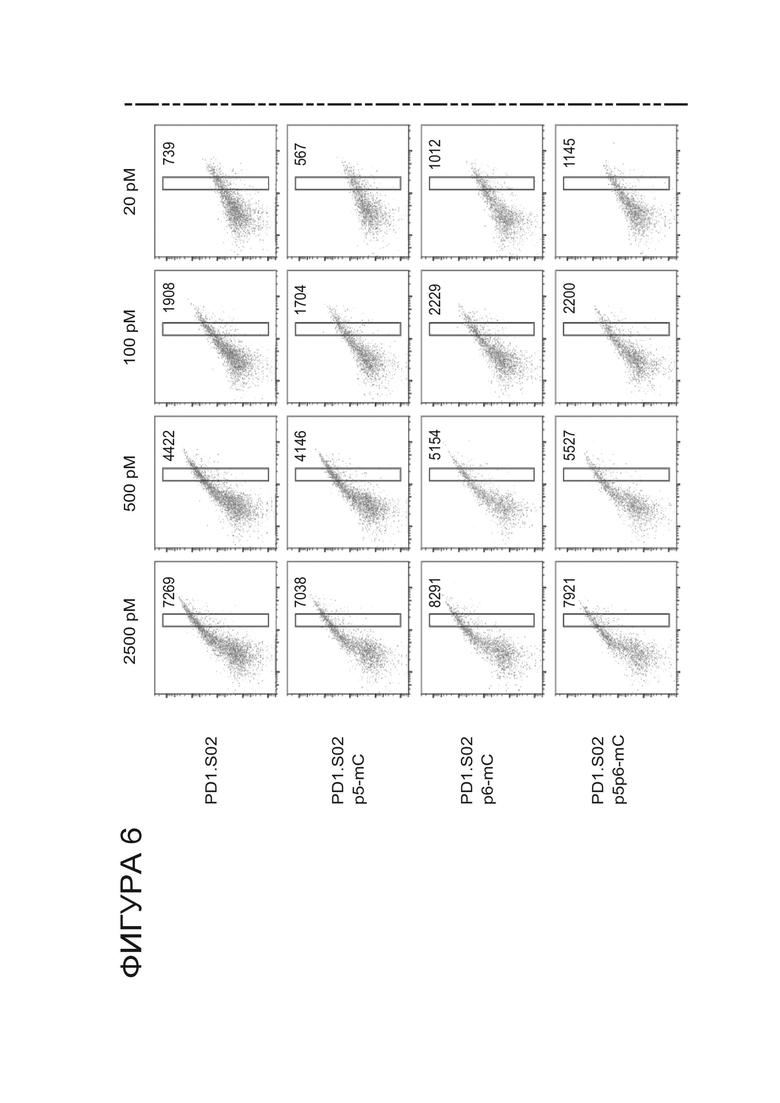


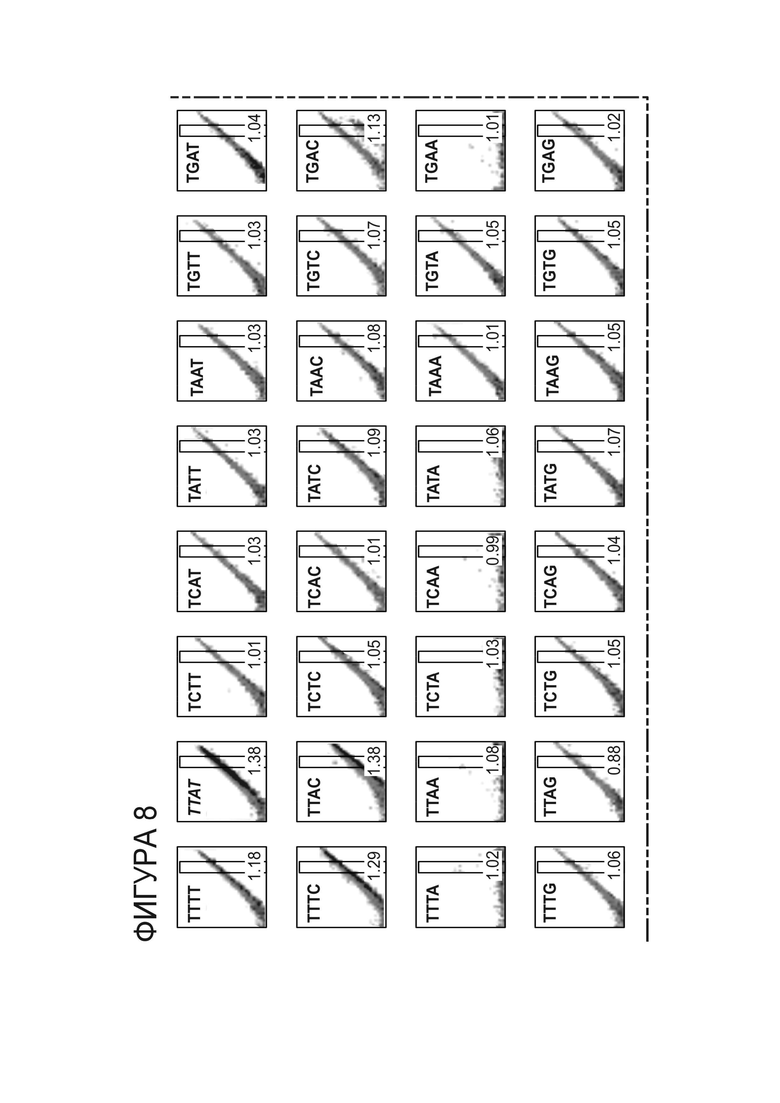







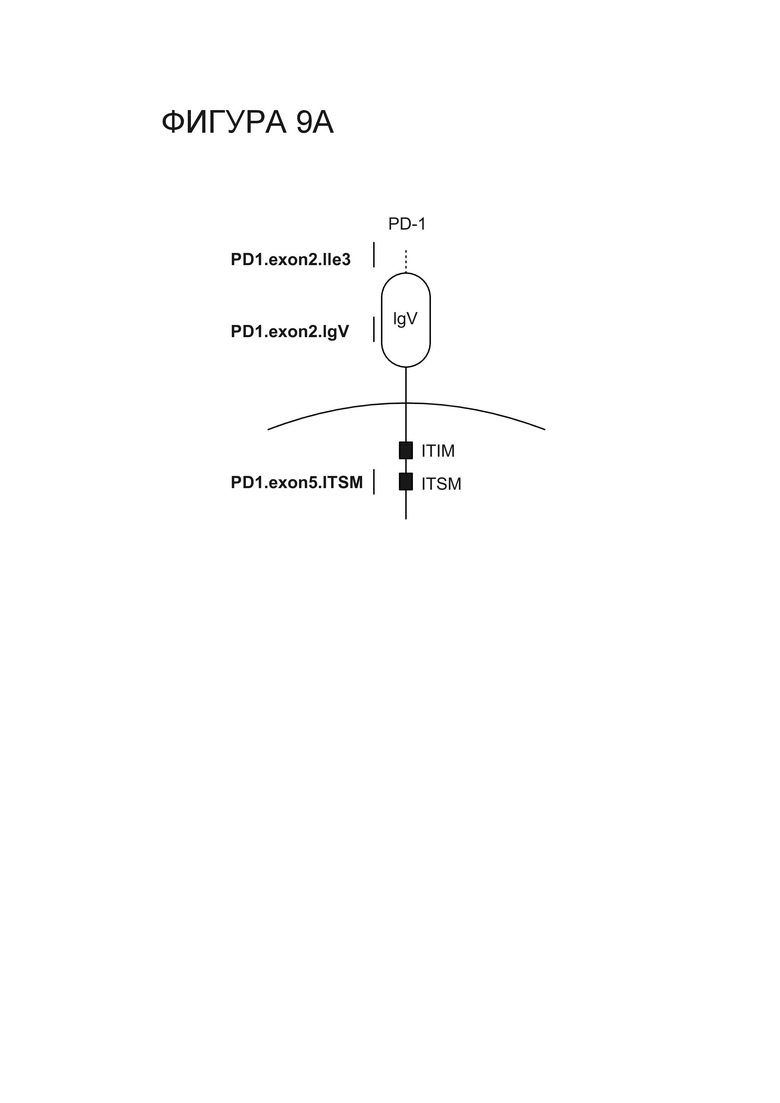

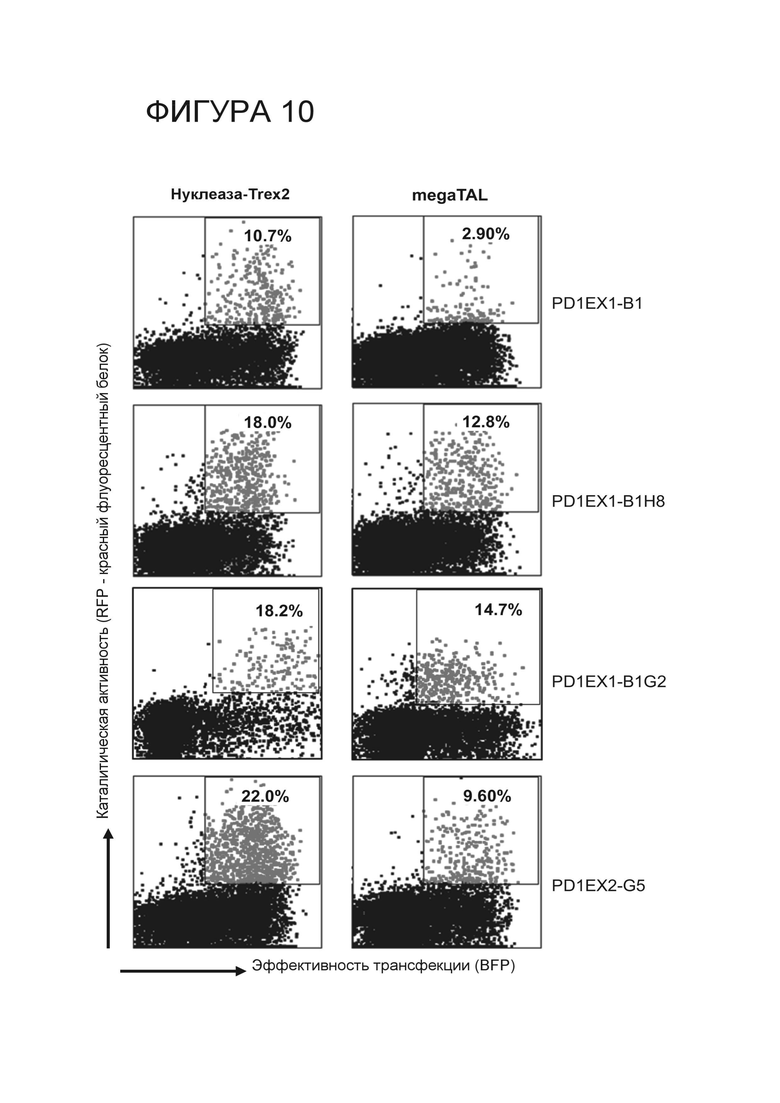

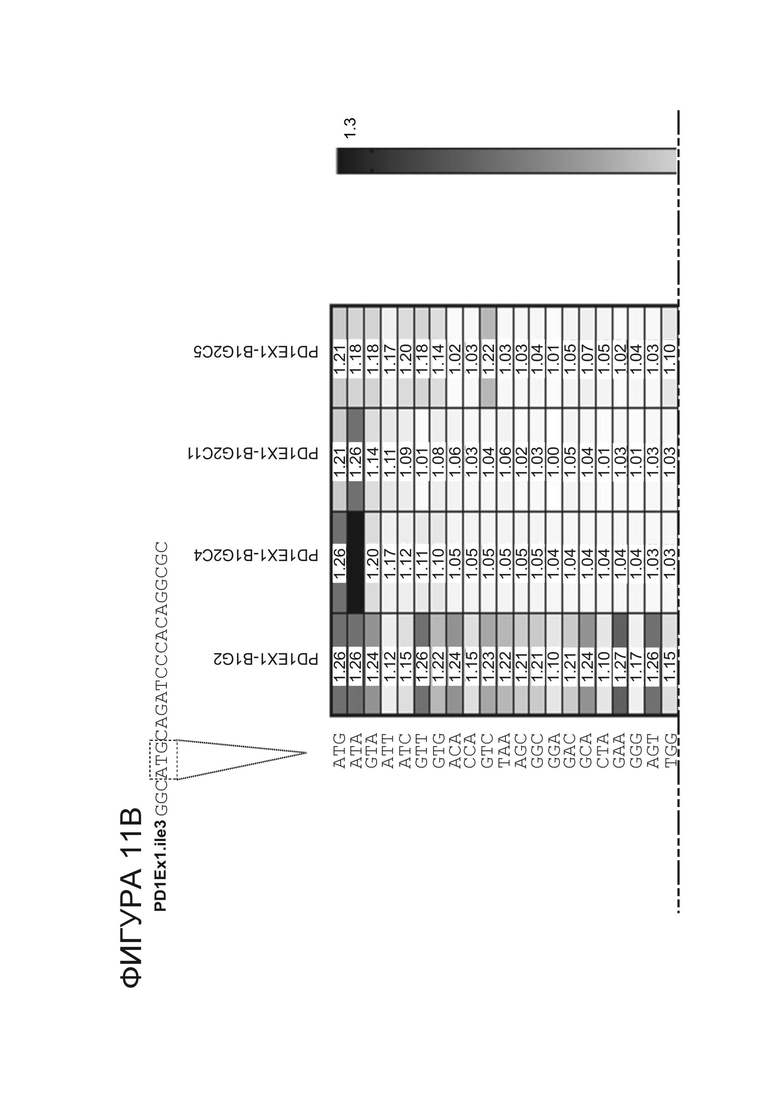


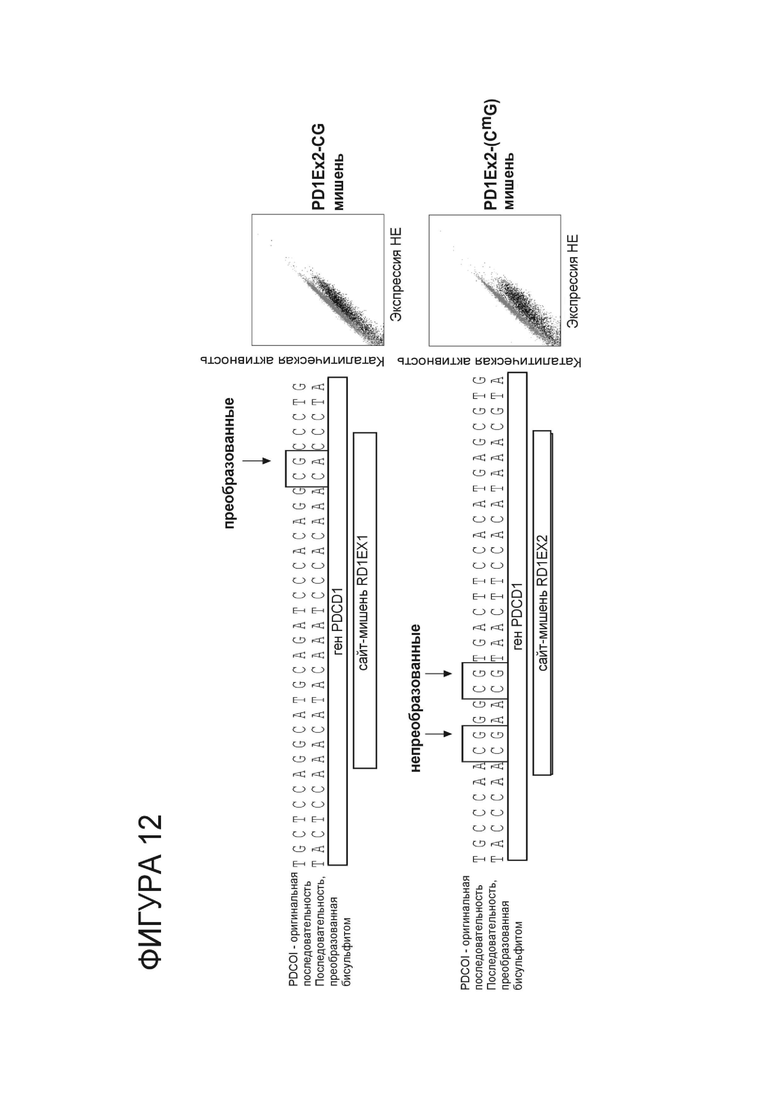
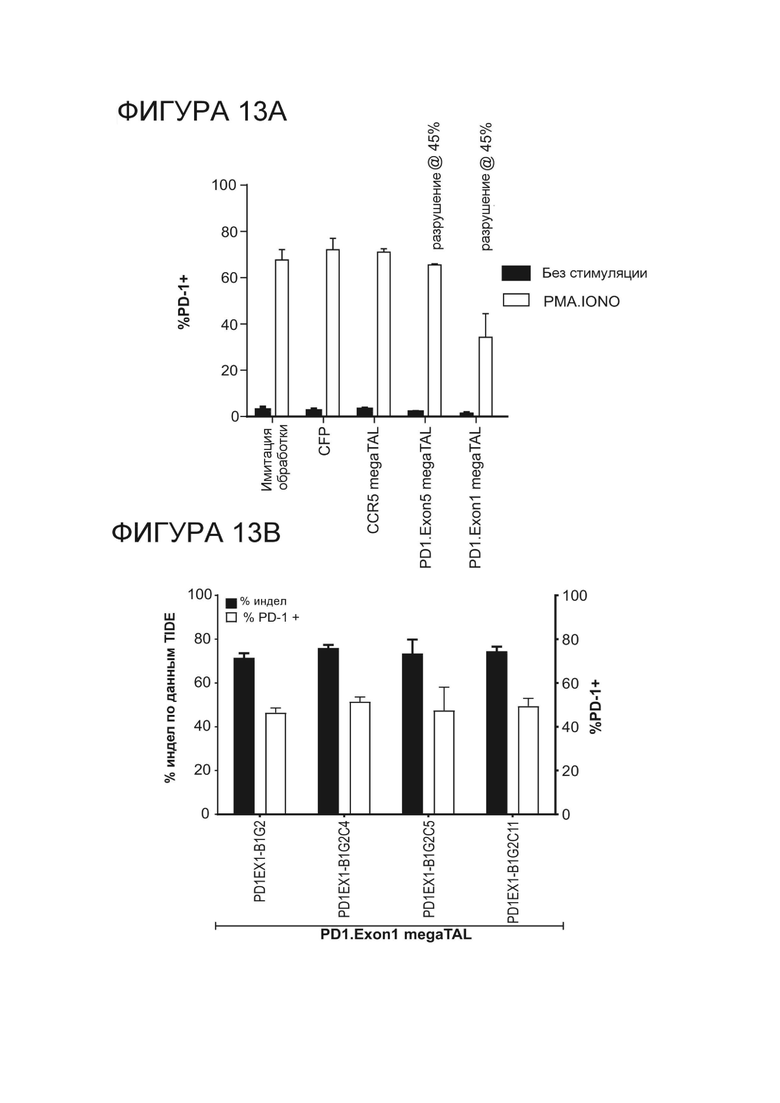
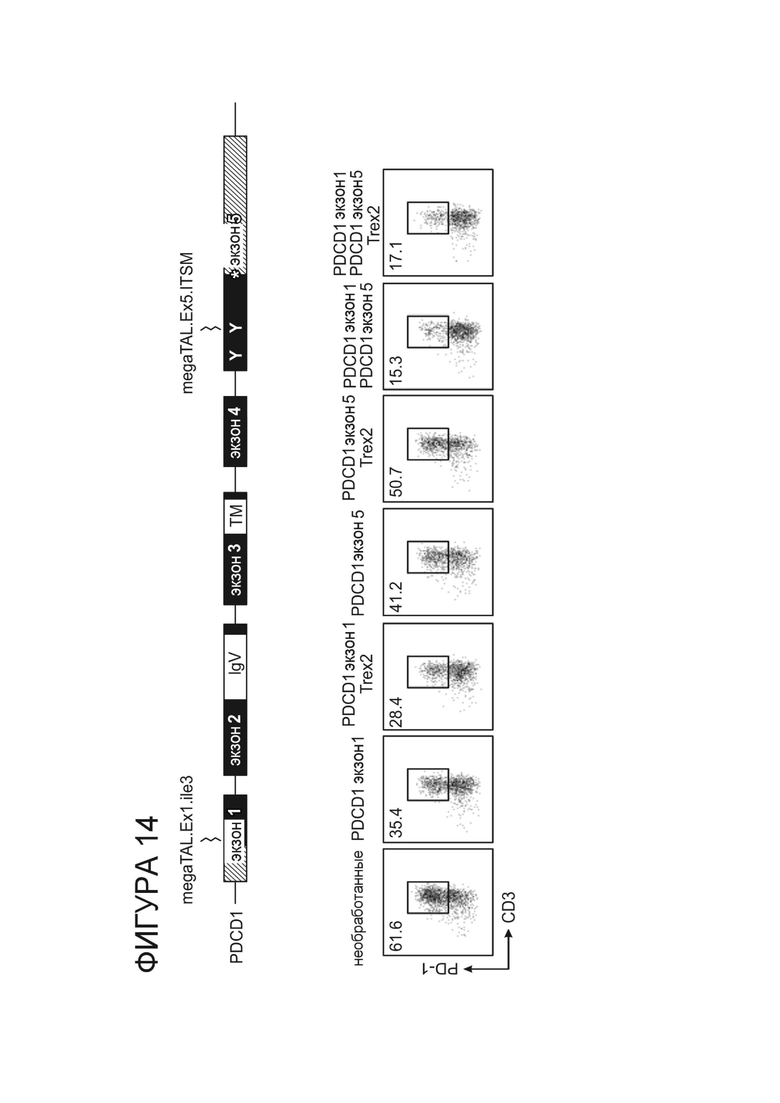
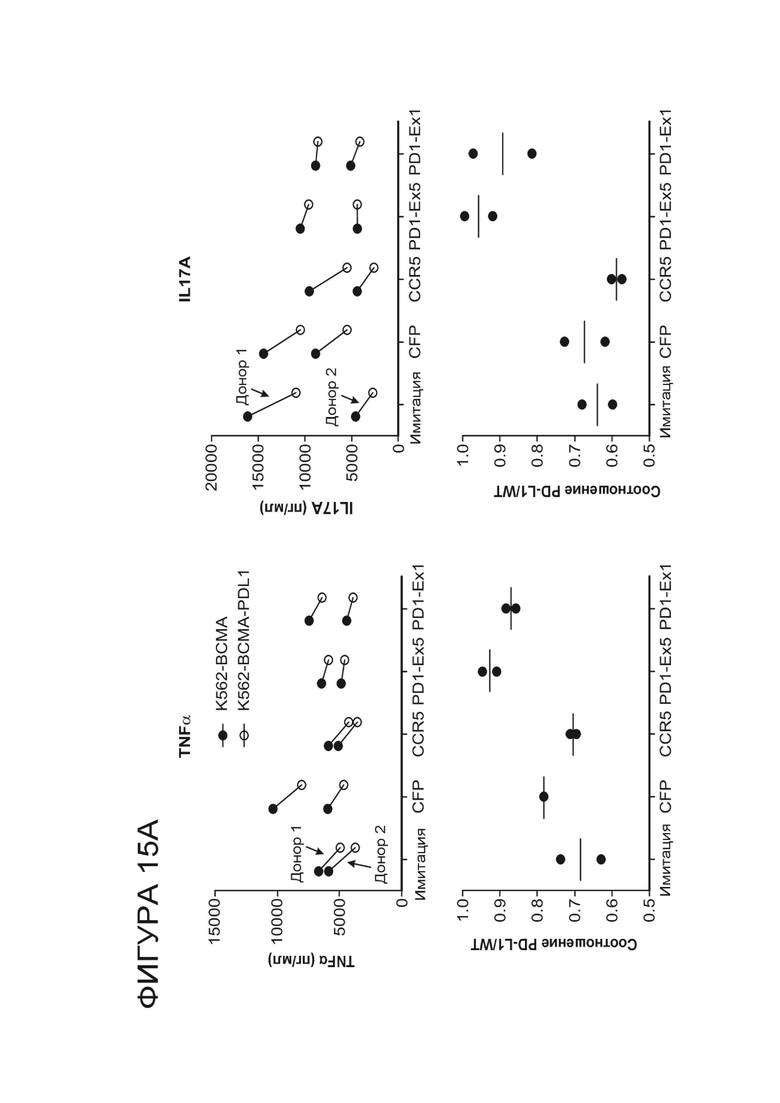
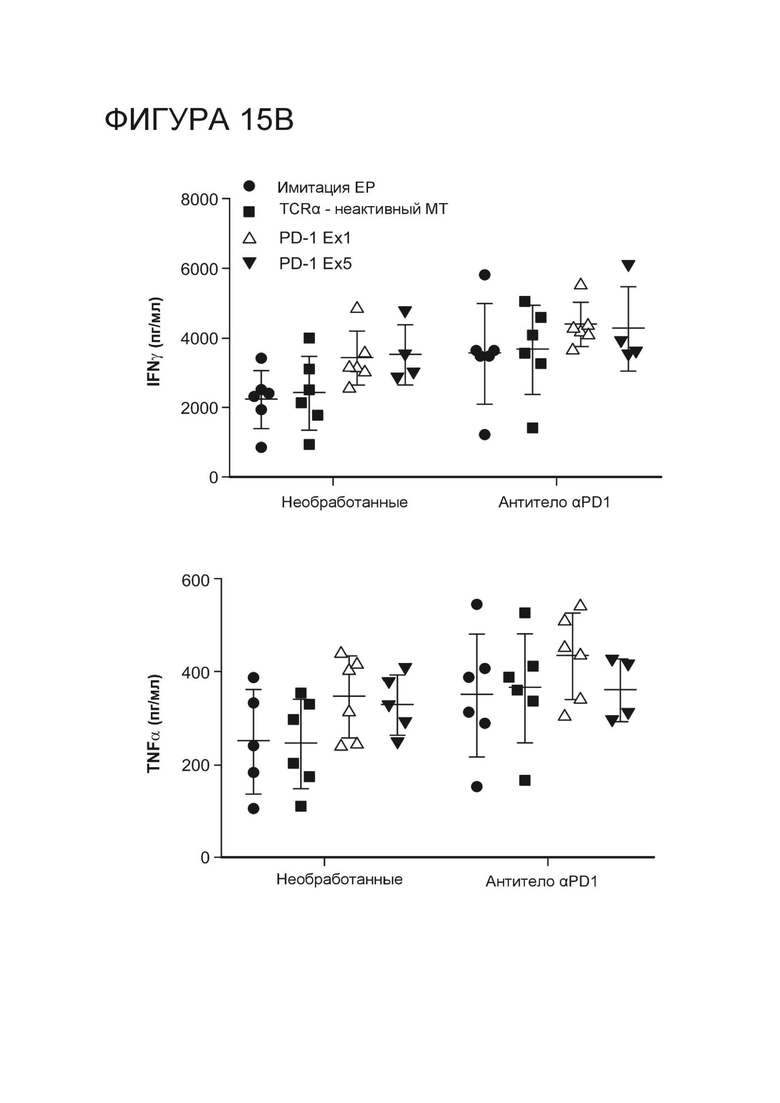
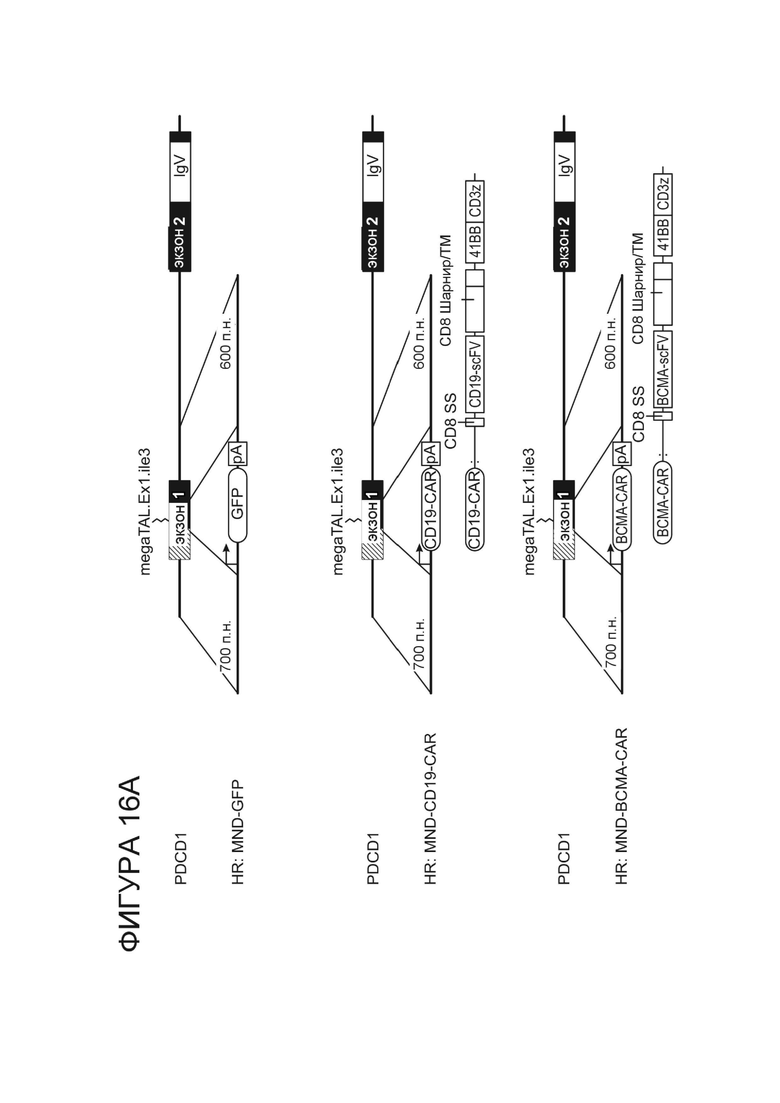
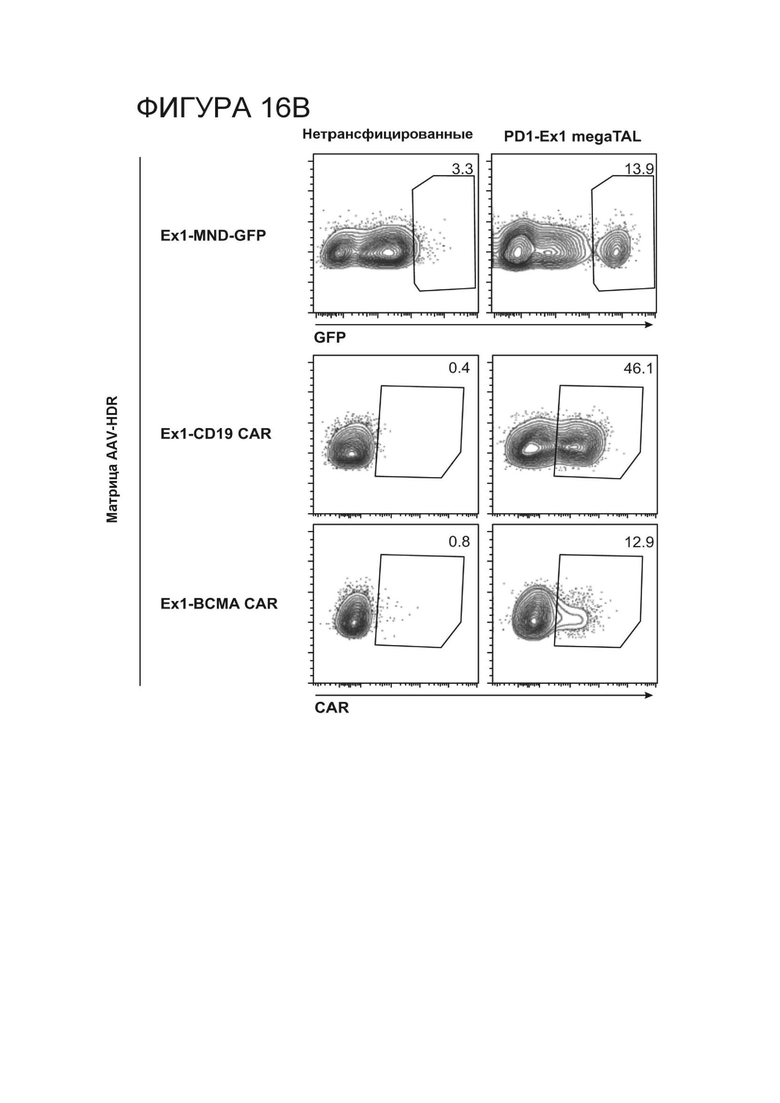
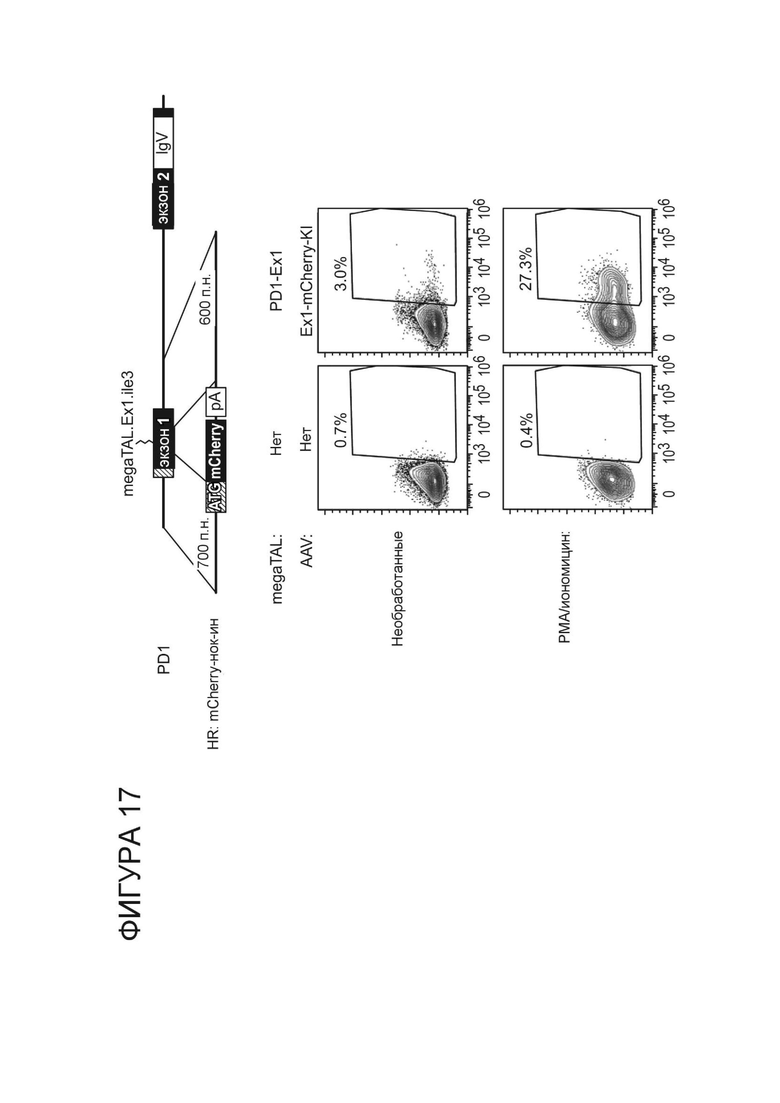
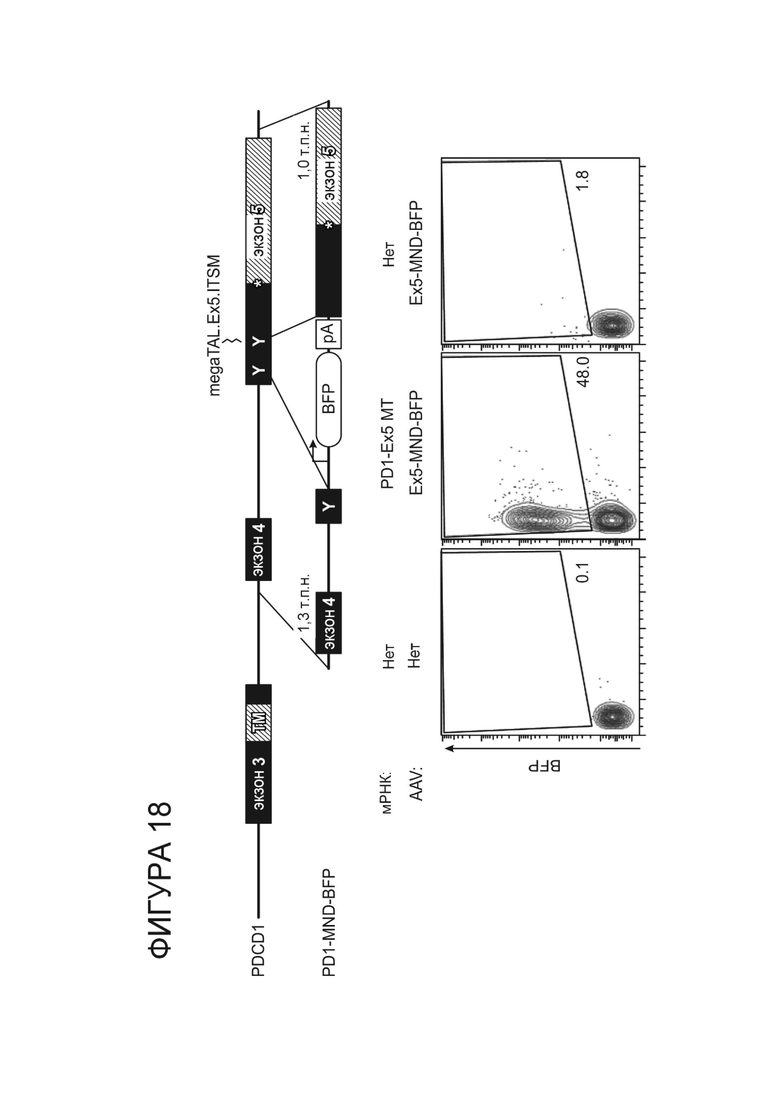
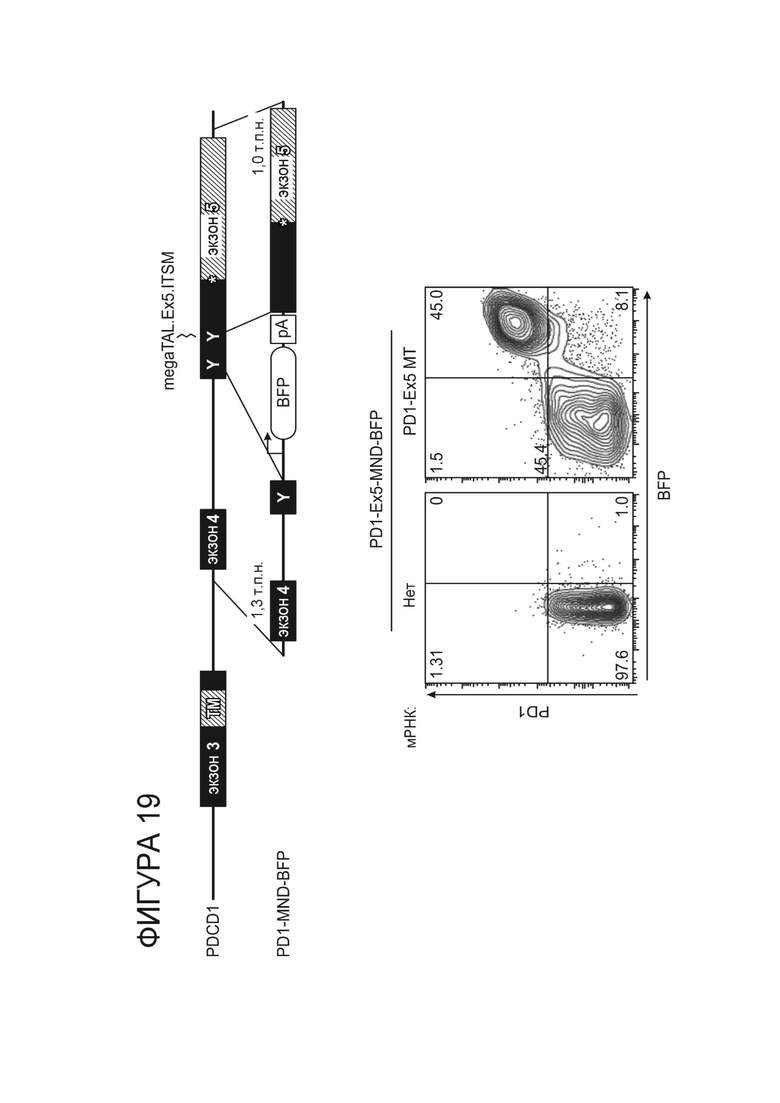
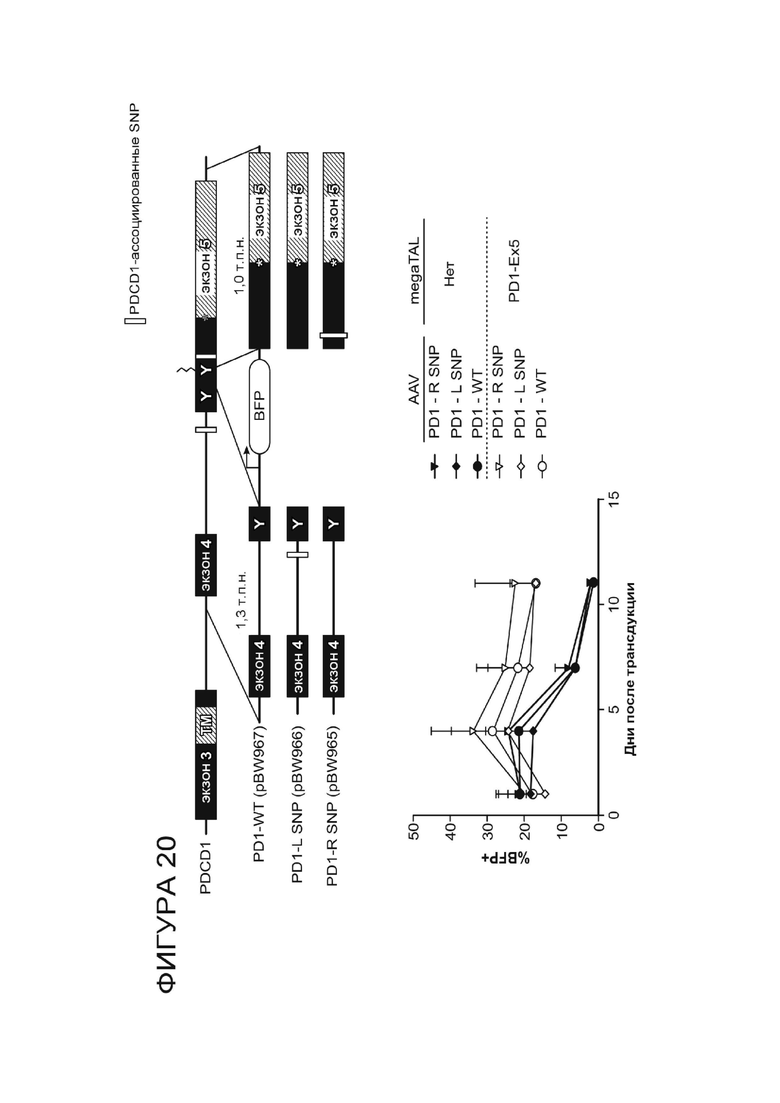
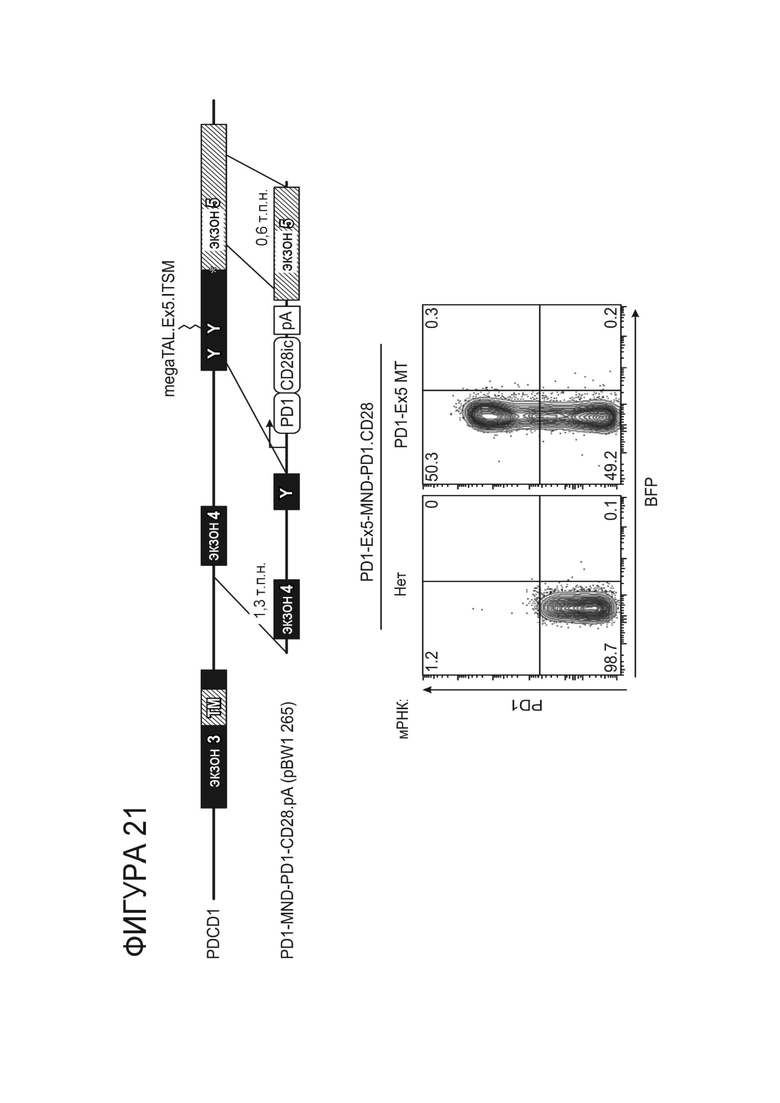
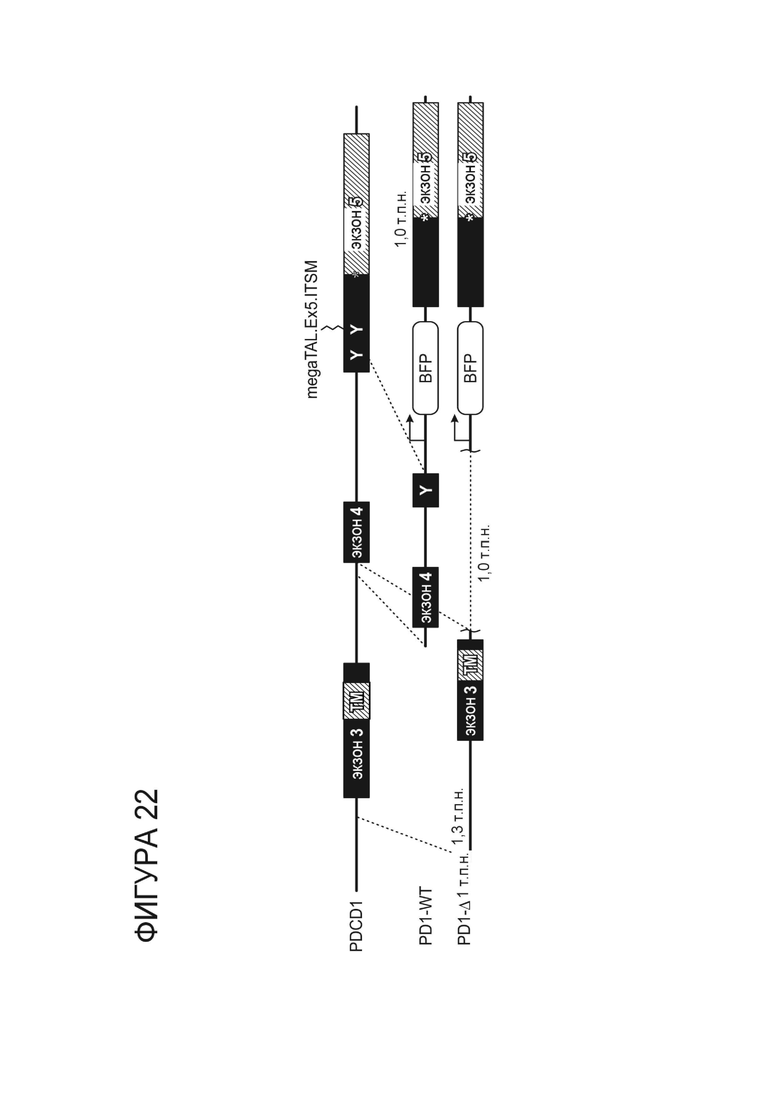
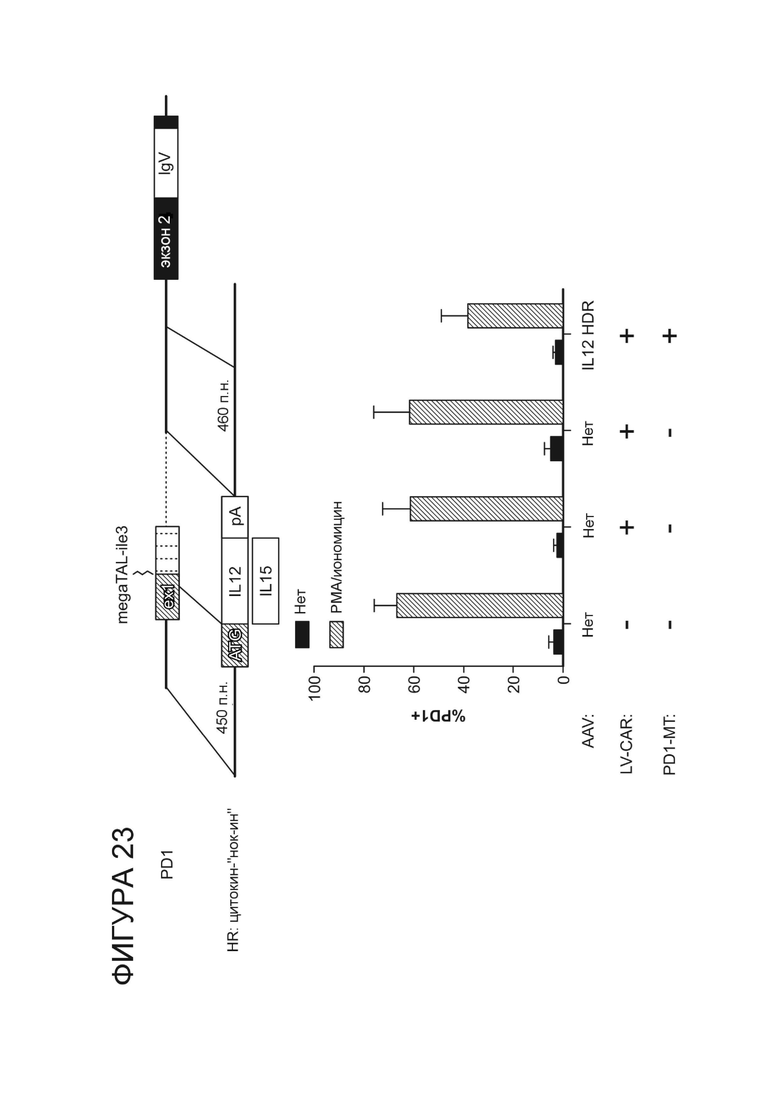
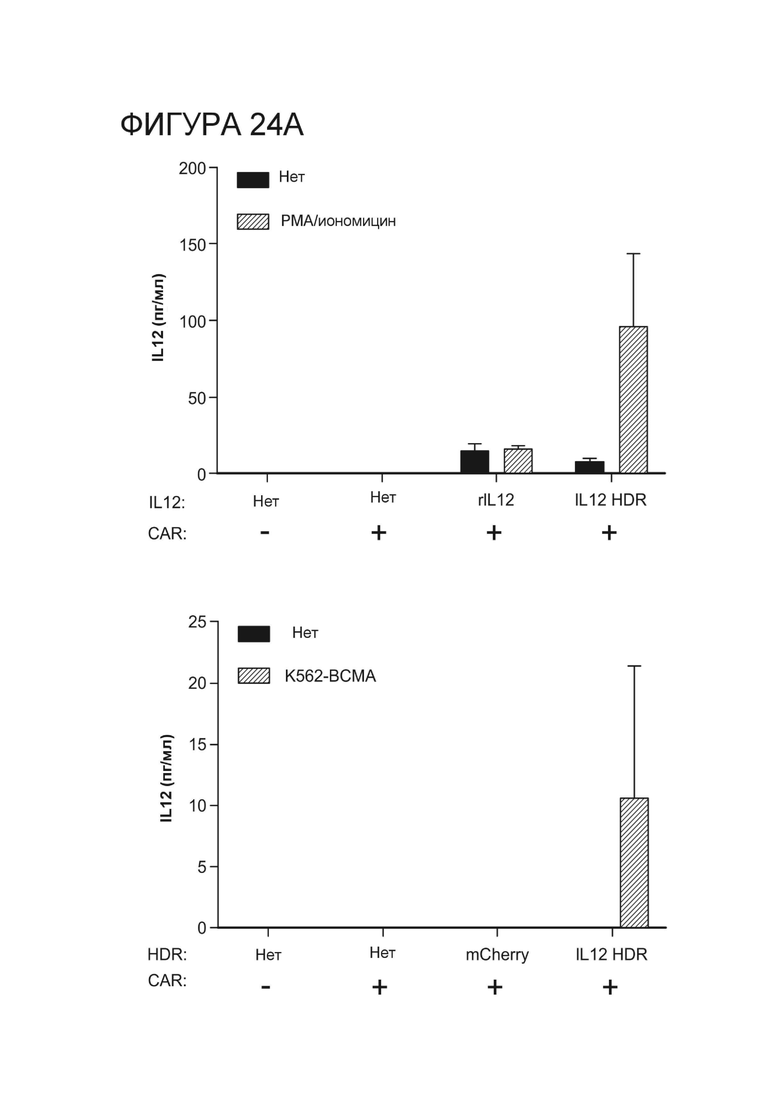

Изобретение относится к области биотехнологии, в частности к полипептиду, содержащему вариант I-OnuI хоминг-эндонуклеазы (НЕ), а также к полинуклеотиду его кодирующему. Также раскрыт вектор, содержащий вышеуказанный полинуклеотид. Изобретение также относится к клетке, содержащей вышеуказанный полипептид или полинуклеотид, а также к композиции, содержащей вышеуказанную клетку. Изобретение эффективно для адоптивной клеточной терапии. 9 н. и 7 з.п. ф-лы, 24 ил., 2 табл., 14 пр.
1. Полипептид, содержащий вариант I-OnuI хоминг-эндонуклеазы (НЕ), который связывает и расщепляет сайт-мишень в человеческом гене запрограммированной клеточной гибели 1 (PD-1), отличающийся тем, что вариант I-OnuI НЕ содержит аминокислотную последовательность, по меньшей мере на 95% идентичную аминокислотной последовательности, представленной в любой из SEQ ID NO: 60-63.
2. Полипептид по п. 1, где вариант I-OnuI НЕ расщепляет сайт-мишень экзона 1 гена PD-1 и содержит следующие аминокислотные замены по отношению к аминокислотной последовательности, представленной в SEQ ID NO: 4:
(a) L26G, R28S, R30L, N32R, K34R, S35G, S36T, V37A, G38R, S40H, E42R, G44S, Q46A, T48V, V68I, А70Т, S72D, N75R, A76Y, S78R, K80R, I100V, L138M, T143N, S159P, E178D, C180S, N184R, I186R, K189N, S190V, K191N, L192A, G193R, Q195R, S201E, T203S, K207R, Y223H, K225Y, K227G, F232R, D236Q, V238R и Т240Е;
(b) L26G, R28S, R30L, N32R, K34R, S35G, S36T, V37A, V37G, G38R, S40H, E42R, G44S, Q46T, T48V, V68I, А70Т, S72D, N75R, A76Y, S78R, К80С, I100V, V132A, L138M, T143N, S155G, S159P, E178D, C180S, N184R, I186R, K189N, S190V, K191N, L192A, G193R, Q195R, S201E, T203S, K207R, Y223H, K225Y, K227G, F232R, D236Q, V238R и Т240Е;
(c) L26G, R28S, R30L, N32R, K34R, S35G, S36T, V37A, G38R, S40H, E42R, G44S, Q46T, Т48М, V68I, А70Т, S72N, N75H, A76Y, S78T, K80R, I100V, L138M, T143N, S159P, E178D, C180S, N184R, I186R, K189N, S190V, K191N, L192A, G193R, Q195R, S201E, T203S, K207R, Y223H, K225Y, K227G, F232R, D236Q, V238R и Т240Е;
(d) L26G, R28S, R30L, N32R, K34R, S35G, S36T, V37A, G38R, S40H, E42R, G44S, Q46T, Т48М, V68S, A70Y, S72N, N75H, A76Y, K80E, T82F, L138M, T143N, S159P, E178D, C180S, N184R, I186R, K189N, S190V, K191N, L192A, G193R, Q195R, S201E, T203S, K207R, Y223H, K225Y, K227G, F232R, D236Q, V238R и Т240Е;
(e) L26G, R28S, R30L, N32R, K34R, S35G, S36T, V37A, G38R, S40H, E42R, G44S, Q46T, Т48М, V68S, A70L, S72N, N75H, A76Y, K80V, T82Y, L138M, T143N, S159P, E178D, C180S, N184R, I186R, K189N, S190V, K191N, L192A, G193R, Q195R, S201E, T203S, K207R, Y223H, K225Y, K227G, F232R, D236Q, V238R и Т240Е; или
(f) L26G, R28S, R30L, N32R, K34R, S35G, S36T, V37G, G38R, S40H, E42R, G44S, Q46T, Т48М, V68S, А70Т, S72N, N75H, A76Y, K80V, T82Y, L138M, T143N, S159P, E178D, C180S, N184R, I186R, K189N, S190V, K191N, L192A, G193R, Q195R, S201E, T203S, K207R, Y223H, K225Y, K227G, F232R, D236Q, V238R и Т240Е.
3. Полипептид по п. 1 или 2, где указанный полипептид связывает полинуклеотидную последовательность, представленную в SEQ ID NO: 30.
4. Полипептид, содержащий вариант I-OnuI хоминг-эндонуклеазы (НЕ), который связывает и расщепляет сайт-мишень в человеческом гене запрограммированной клеточной гибели 1 (PD-1), отличающийся тем, что вариант I-OnuI НЕ содержит аминокислотную последовательность, по меньшей мере на 95% идентичную аминокислотной последовательности, представленной в любой из SEQ ID NO: 60-63, дополнительно содержащий ДНК-связывающий домен TALE, который содержит от приблизительно 9,5 единиц повтора TALE до приблизительно 15,5 единиц повтора TALE.
5. Полипептид по п. 4, где ДНК-связывающий домен TALE связывает полинуклеотидную последовательность, представленную в SEQ ID NO: 31.
6. Полипептид по п. 5, где указанный полипептид связывает и расщепляет полинуклеотидную последовательность, представленную в SEQ ID NO: 32.
7. Полипептид, содержащий вариант I-OnuI хоминг-эндонуклеазы (НЕ), который связывает и расщепляет сайт-мишень в человеческом гене запрограммированной клеточной гибели 1 (PD-1), отличающийся тем, что вариант I-OnuI НЕ содержит аминокислотную последовательность, по меньшей мере на 95% идентичную аминокислотной последовательности, представленной в любой из SEQ ID NO: 60-63, дополнительно содержащий пептидный линкер или вирусный саморасщепляемый пептид 2А и 3'-5' экзонуклеазу Trex2, или соответствующий биологически активный фрагмент.
8. Полинуклеотид, кодирующий полипептид по любому из пп. 1-7.
9. Полинуклеотид по п. 8, где полинуклеотид представляет собой мРНК.
10. Полинуклеотид по п. 8, где полинуклеотид представляет собой кДНК.
11. Вектор экспрессии, содержащий полинуклеотид, кодирующий полипептид по любому из пп. 1-10.
12. Клетка для адоптивной клеточной терапии, содержащая полипептид по любому из пп. 1-7, полинуклеотид по пп. 8-10 или вектор экспрессии по п. 11, при этом клетка представляет собой гемопоэтическую клетку, Т-клетку, клетку CD3+, клетку CD4+, клетку CD8+, иммунную эффекторную клетку, цитотоксический Т-лимфоцит (ЦТЛ), инфильтрирующий опухоль лимфоцит (TIL), хелперную Т-клетку, природный киллер (NK) или Т-клетку-природный киллер (NKT).
13. Клетка для адоптивной клеточной терапии, содержащая полипептид по любому из пп. 1-7, полинуклеотид по любому из пп. 8-10 или вектор экспрессии по п. 11, где указанная клетка содержит полинуклеотид, кодирующий сконструированный TCR, CAR, рецептор Daric или дзетакин.
14. Клетка для адоптивной клеточной терапии по п. 13, где указанная клетка представляет собой гемопоэтическую клетку, Т-клетку, CD3+ клетку, CD4+ клетку, CD8+ клетку, иммунную эффекторную клетку, цитотоксический Т-лимфоцит (CTL), инфильтрирующий опухоль лимфоцит (TIL), хелперную Т-клетку, клетку-природный киллер (NK) или Т-клетку-природный киллер (NKT).
15. Композиция для адоптивной клеточной терапии, содержащая эффективное количество клеток по любому из пп. 12-14.
16. Композиция для адоптивной клеточной терапии, содержащая эффективное количество клеток по любому из пп. 12-14 и физиологически приемлемый носитель.
| EA201492222 А1, 29.05.2015 | |||
| WO2012068380 A2, 24.05.2012 | |||
| WO2007049095 A1, 03.05.2007 | |||
| УПРАВЛЯЮЩЕЕ УСТРОЙСТВО ДЛЯ ПРЕДОХРАНИТЕЛЬНОГО КЛАПАНА | 1999 |
|
RU2215223C2 |
| WO2013126794 A1, 29.08.2013 | |||
| ЖДАНОВ Д.Д | |||
| и др | |||
| Эндонуклеаза EndoG индуцирует альтернативный сплайсинг каталитической субъединицы теломеразы hTERT и гибель опухолевых клеток, Биомедицинская химия, 2016, том 62, вып | |||
| Переносная печь для варки пищи и отопления в окопах, походных помещениях и т.п. | 1921 |
|
SU3A1 |
| Коловратный насос с кольцевым поршнем, перемещаемым эксцентриком | 1921 |
|
SU239A1 |
Авторы
Даты
2022-10-05—Публикация
2017-09-08—Подача